РОДСТВЕННЫЕ ЗАЯВКИ
Настоящая заявка испрашивает приоритет на основании предварительной заявки на патент США №62/268158, поданной 16 декабря 2015 г., содержание которой включено в данный документ посредством ссылки.
ЗАЯВЛЕНИЕ ОБ ЭЛЕКТРОННОЙ ПОДАЧЕ ПЕРЕЧНЯ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
Предоставляется перечень последовательностей в текстовом формате ASCII, под названием 80955 SEQ LIST_ST25.txt и размером 122 килобайт, созданный 5 декабря 2016 г., и электронный перечень последовательностей подается совместно с настоящей заявкой. Данный перечень последовательностей тем самым включен посредством ссылки в описание данного документа для его раскрытия.
ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящее изобретение относится к композициям и способам для введения в растение аллелей, генов и/или хромосомных интервалов, которые обеспечивают у указанного растения признаки повышенной засухоустойчивости, и/или повышенной урожайности в условиях недостатка воды, и/или повышенной урожайности в отсутствие недостатка воды.
ПРЕДПОСЫЛКИ ИЗОБРЕТЕНИЯ
Засуха является одним из главных ограничений для производства маиса по всему миру. Ежегодные потери мировой сельскохозяйственной культуры маиса вследствие засухи составляют около 15%. Периоды стресса, вызванного засухой, могут наблюдаться в любой момент времени в течение периода вегетации. Маис является особенно чувствительным к стрессу, вызванному засухой, в период времени непосредственно перед цветением и в течение него. Когда стресс, вызванный засухой, наблюдается в течение данного критического периода, это может приводить к значительному снижению урожайности зерна.
Идентификация генов, которые улучшают засухоустойчивость сельскохозяйственных культур, могла бы привести к более эффективным методам производства сельскохозяйственных культур путем обеспечения идентификации, отбора и производства культурных растений с повышенной засухоустойчивостью.
Соответственно, целью селекции растений является объединение разных требуемых признаков в одном растении. Для полевых культур, таких как кукуруза, соя и т.д., эти признаки могут включать большую урожайность и лучшее агрономическое качество. Однако генетические локусы, которые воздействуют на урожайность и агрономическое качество, не всегда известны, а даже если они известны, их вклад в такие признаки зачастую является не ясным. Таким образом, необходимо идентифицировать новые локусы, которые могут позитивно воздействовать на такие требуемые признаки, и/или необходимо исследовать возможности известных локусов по такому воздействию.
После обнаружения эти требуемые локусы можно подвергать отбору в качестве части селекционной программы для получения растений, которые несут требуемые признаки. Иллюстративный вариант осуществления способа получения таких растений включает перенос последовательностей нуклеиновой кислоты за счет интрогрессии из растений, которые имеют требуемую генетическую информацию, в растения, которые ее не имеют, путем скрещивания растений с применением традиционных методик селекции. Кроме того, можно применять недавно открытые возможности редактирования генома для редактирования генома растения так, чтобы он содержал требуемые гены или аллельные формы генов.
Требуемые локусы можно вводить в коммерчески доступные сорта растений с применением отбора с помощью маркеров (MAS), селекции с помощью маркеров (МАВ), трансгенной экспрессии гена(-ов) и/или за счет современных технологий редактирования генов, таких как, например, CRISPR, TALEN и т.д.
Поэтому необходимы новые способы и композиции для введения в растение гена или участка генома, которые могут приводить к получению засухоустойчивых сельскохозяйственных культур и/или сельскохозяйственных культур, которые характеризуются повышенной урожайностью как в условиях достаточного наличия воды, так и в условиях недостатка воды.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
В данном кратком описании перечислены некоторые варианты осуществления объектов, раскрытых в настоящем изобретении, и для многих случаев перечислены вариации и преобразования этих вариантов осуществления. Данное краткое описание изобретения является всего лишь иллюстрацией многочисленных и разнообразных вариантов осуществления. Упоминание одного или нескольких репрезентативных признаков рассматриваемого варианта осуществления также является иллюстративным. Такой вариант осуществления, как правило, может встречаться с упомянутым(-и) признаком(-ами) или без него(них); аналогично эти признаки могут применяться к другим вариантам осуществления объекта, раскрытого в настоящем изобретении, независимо от того, перечислены они в кратком описании или нет. Чтобы избежать избыточного повторения, в данном кратком описании не перечислены или не предполагаются все возможные комбинации таких признаков.
Предусмотрены композиции и способы для идентификации, отбора и/или получения растений с повышенной урожайностью в условиях засухи. Как описано в данном документе, участки генома (взаимозаменяемо - "хромосомные интервалы") могут содержать, состоять фактически или состоять из гена(ов), одного аллеля или комбинации аллелей в одном или нескольких генетических локусах, ассоциированных с повышенной засухоустойчивостью и/или повышенной урожайностью.
Все положения в хромосомах маиса, раскрытые в данном документе, соответствуют "эталонному геному В73, версия 2" маиса. "Эталонный геном В73, версия 2" представляет собой имеющиеся в открытом доступе физический и генетический остов генома маиса В73. Он является результатом программы работ по секвенированию с использованием минимальной дорожки перекрывания из примерно 19000 картированных клонов ВАС, и нацелен на получение высококачественного покрытия последовательностей всех идентифицируемых ген-содержащих участков генома маиса. Эти участки были упорядочены, ориентированы и вместе со всеми межгенными последовательностями закреплены в существующих физических и генетических картах генома маиса. Доступ к нему можно получить с применением программы для просмотра генома, Maize Genome Browser, находящейся в открытом доступе в сети Интернет, которая может облегчить пользователю взаимодействие с последовательностями и данными карты.
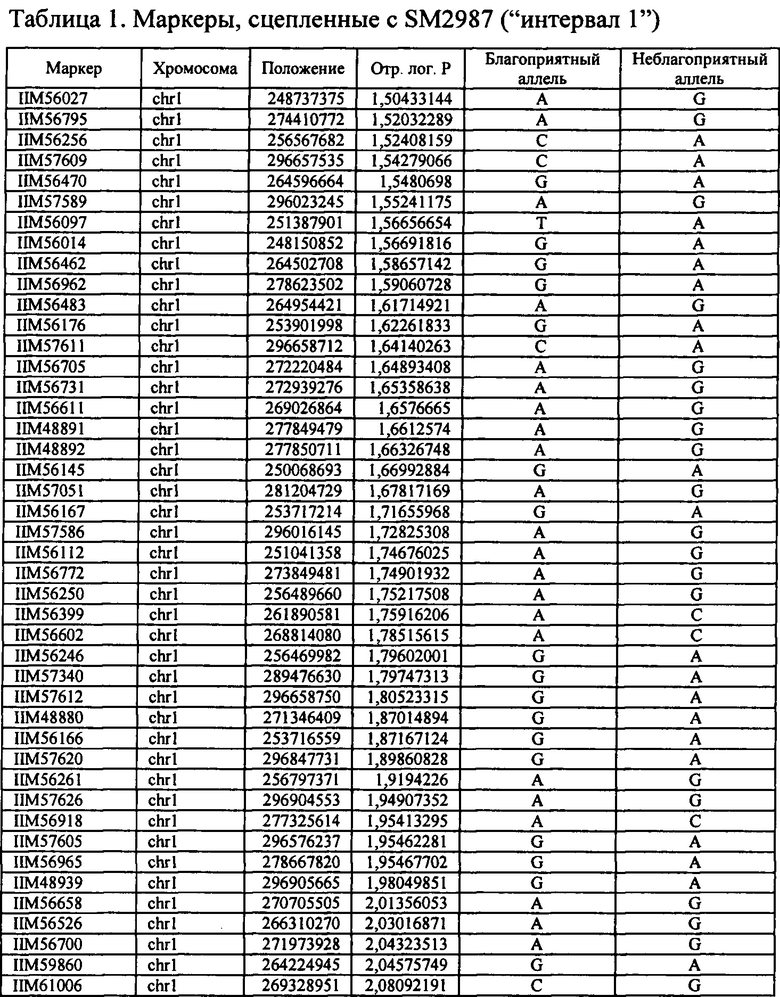
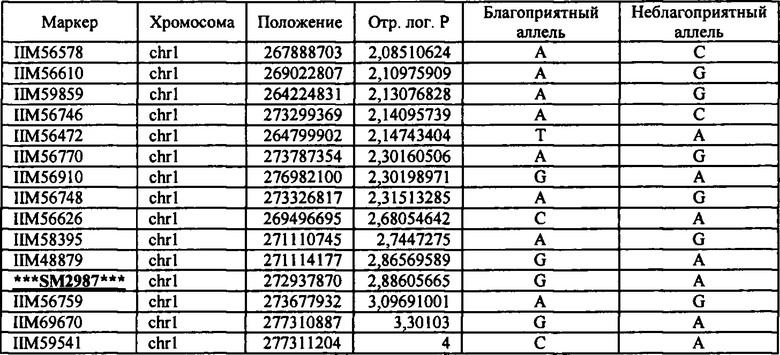
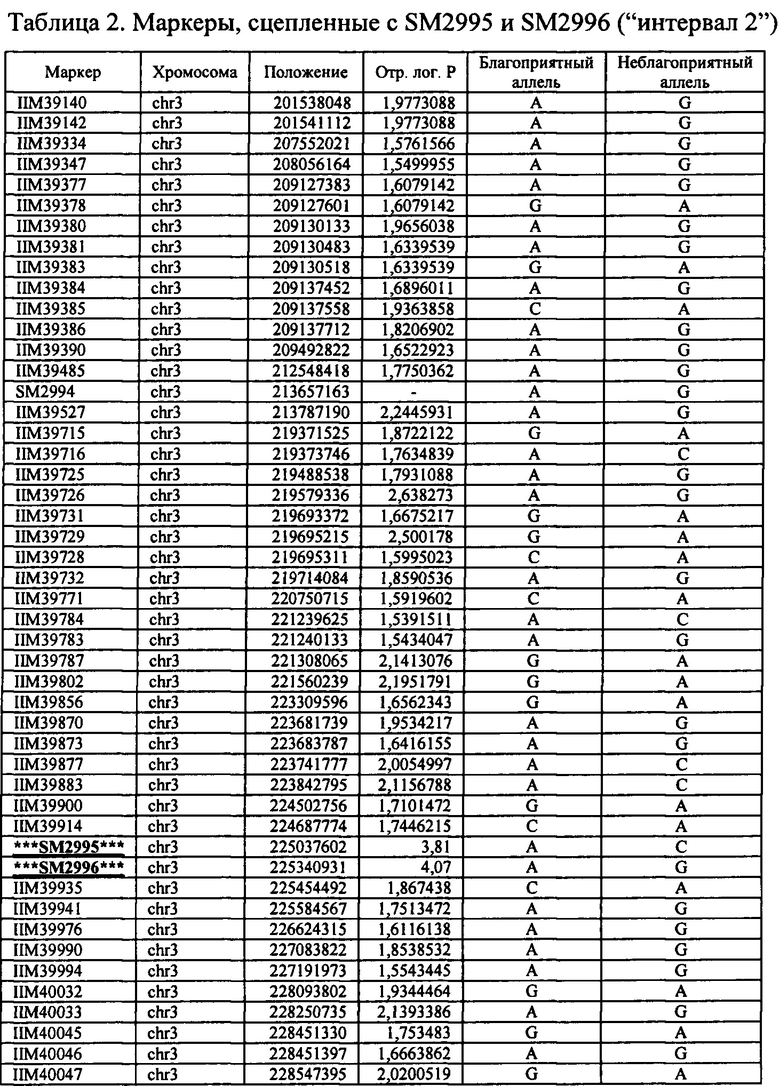
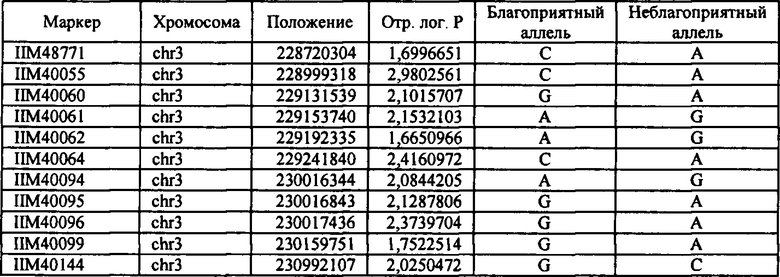
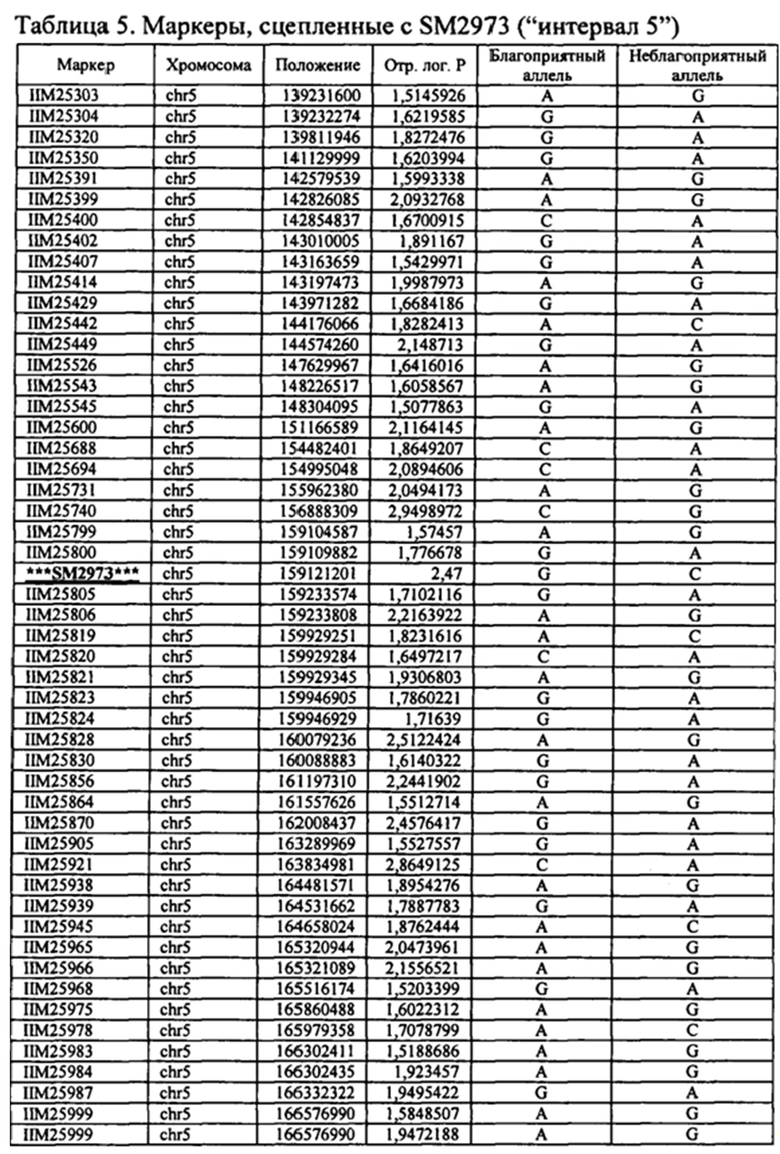
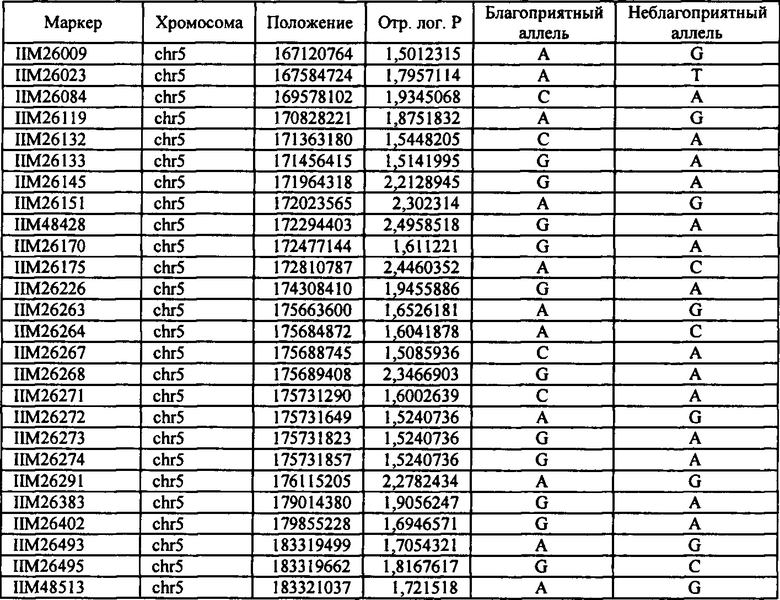
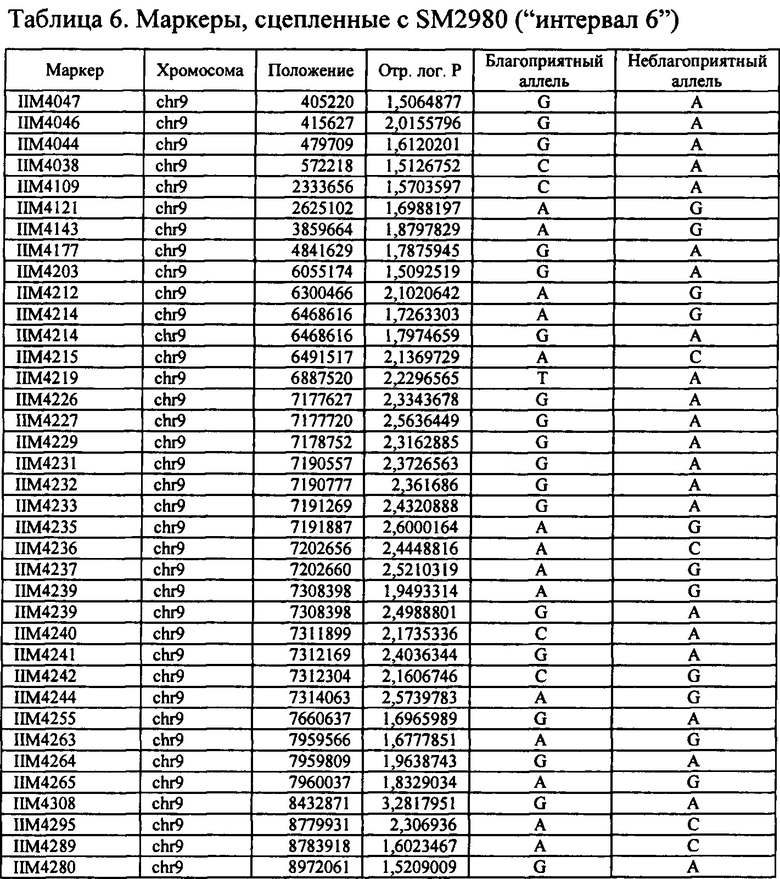
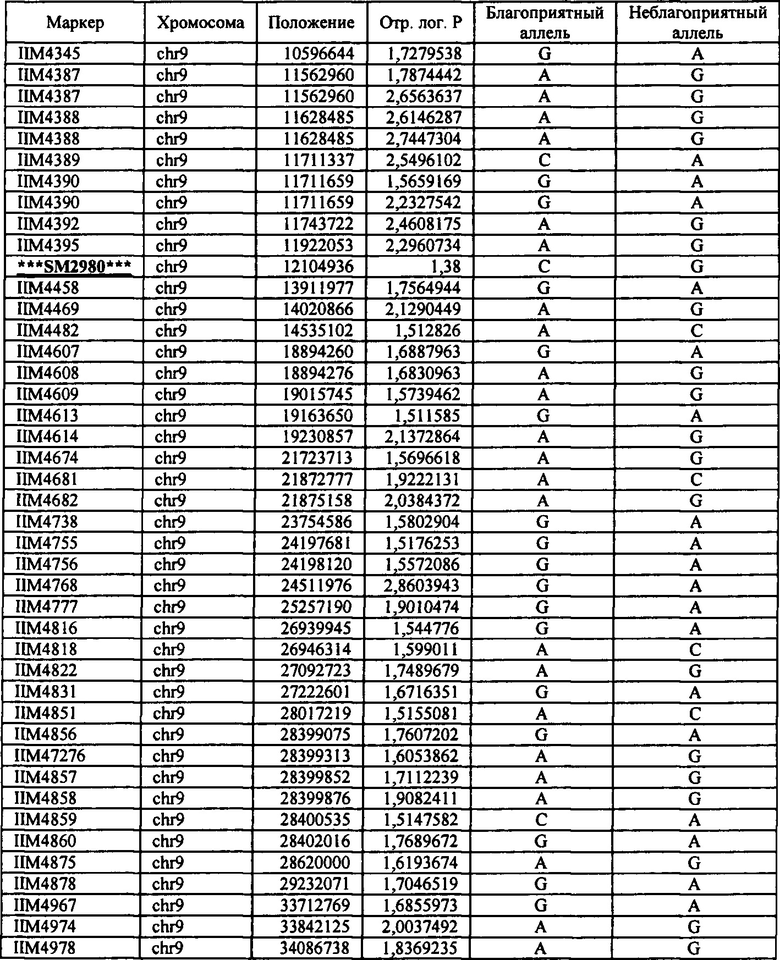
В настоящем изобретении в пределах генома маиса были идентифицированы восемь причинных локусов, которые тесно ассоциированы с повышенной засухоустойчивостью (например, увеличением количества бушелей кукурузы на акр в условиях засухи) и с повышенной урожайностью (например, увеличением количества бушелей кукурузы на акр в условиях, отличных от засухи, нормальных условиях или условиях достаточного наличия воды), причем эти восемь локусов совместно обозначены в данном документе как (аллели урожайности'). В частности, в настоящем изобретении раскрыты следующие восемь аллелей урожайности, которые разграничивают центральные тесно ассоциированные с урожайностью локусы, причем эти аллели включают: (1) SM2987 (в данном документе ('аллель урожайности 1') или ('SM2987')), расположенный на хромосоме 1 маиса, который соответствует аллелю G в положении 272937870; (2) SM2991 (в данном документе ('аллель урожайности 2') или ('SM2991')), расположенный на хромосоме 2 маиса, который соответствует аллелю G в положении 12023706; (3) SM2995 (в данном документе ('аллель урожайности 3') или ('SM2995')), расположенный на хромосоме 3 маиса, который соответствует аллелю А в положении 225037602; (4) SM2996 (в данном документе ('аллель урожайности 4') или ('SM2996')), расположенный на хромосоме 3 маиса, который соответствует аллелю А в положении 225340931; (5) SM2973 (в данном документе ('аллель урожайности 5') или ('SM2973')), расположенный на хромосоме 5 маиса, который соответствует аллелю G в положении 159121201; (6) SM2980 (в данном документе ('аллель урожайности 6') или ('SM2980')), расположенный на хромосоме 9 маиса, который соответствует аллелю С в положении 12104936; (7) SM2982 (в данном документе ('аллель урожайности 7') или ('SM2982')), расположенный на хромосоме 9 маиса, который соответствует аллелю А в положении 133887717; и (8) SM2984 (в данном документе ('аллель урожайности 8') или ('SM2984')), расположенный на хромосоме 10 маиса, который соответствует аллелю G в положении 4987333 (см. таблицы 1-7). Не ограничиваясь какой-либо теорией, полагают, что каждый из этих аллелей урожайности находится в пределах или возле гена(-ов), который обуславливает рассматриваемый фенотип (например, урожайность как в условиях засухи, так и в условиях, отличных от засухи). Из уровня техники хорошо известно, что маркеры в пределах причинных генов и все тесно ассоциированные маркеры могут применяться в селекции с помощью маркеров для отбора, идентификации и содействия в получении растений, имеющих признак, ассоциированный с рассматриваемым маркером (например, в данном случае, с повышенной засухоустойчивостью и/или урожайностью, см. таблицы 1-7, в которых показаны аллели урожайности и примеры тесно ассоциированных маркеров, которые можно применять для идентификации или получения линий маиса, характеризующихся повышенной засухоустойчивостью, для каждого соответствующего локуса или хромосомного интервала). Соответственно, в одном аспекте настоящего изобретения раскрыт способ отбора или идентификации линии или идиоплазмы маиса, характеризующихся повышенной засухоустойчивостью и/или повышенной урожайностью (т.е. увеличением количества бушелей на акр по сравнению с контрольными растениями), где способ предусматривает стадии: (а) выделения нуклеиновой кислоты из части растения маиса; (b) выявления в нуклеиновой кислоте из (а) молекулярного маркера, который ассоциирован с засухоустойчивостью и/или повышенной урожайностью, где молекулярный маркер является тесно ассоциированным с любым из "аллелей урожайности 1-8", где тесно ассоциированный означает, что маркер находится в пределах 50 сМ, 40 сМ, 30 сМ, 20 сМ, 15 сМ, 10 сМ, 9 сМ, 8 сМ, 7 сМ, 6 сМ, 5 сМ, 4 сМ, 3 сМ, 2 сМ, 1 сМ или 0,5 сМ от указанного аллеля урожайности; и (с) отбора или идентификации растения маиса на основании присутствия указанного маркера на (b). В некоторых вариантах осуществления выбранный маркер из (b) представляет собой любой маркер или тесно ассоциированный маркер, описанные в таблицах 1-7. В других вариантах осуществления маркер из (b) можно применять для получения растений маиса, характеризующихся повышенной засухоустойчивостью или повышенной урожайностью, путем отбора растения маиса в соответствии со способом, описанным в стадиях (а) - (с) выше и дополнительно предусматривающим стадию (d) скрещивания растения из (с) со вторым растением маиса, не содержащим маркер, идентифицированный на (b); и (d) получения растения-потомка, содержащего в своем геноме маркер из (b), где указанное растение-потомок характеризуется повышенной засухоустойчивостью и/или урожайностью по сравнению с контрольным растением. В другом случае также может потребоваться применить тот же маркер, идентифицированный на (b), для отбора растений-потомков, полученных на (d).
В некоторых вариантах осуществления настоящего изобретения представлен способ идентификации и/или отбора засухоустойчивого растения маиса, идиоплазмы маиса или части его растения, причем способ предусматривает: выявление в указанном растении маиса, идиоплазме маиса или части его растения по меньшей мере одного аллеля маркерного локуса, который ассоциирован с засухоустойчивостью у маиса, где указанный по меньшей мере один маркерный локус расположен в пределах хромосомного интервала, выбранного из группы, состоящей из: хромосомного интервала, фланкированного и включающего маркеры IIM56014 и IIM48939 в физических положениях 248150852-296905665 хромосомы 1 (в данном документе "интервал 1"), IIM39140 и IIM40144 в физических положениях 201538048-230992107 хромосомы 3 (в данном документе "интервал 2"), IIM6931 и IIM7657 в физических положениях 121587239-145891243 хромосомы 9 (в данном документе "интервал 3"), IIM40272 и IIM41535 в физических положениях 1317414-36929703 хромосомы 2 (в данном документе "интервал 4"), IIM25303 и IIM48513 в физических положениях 139231600-183321037 хромосомы 5 (в данном документе "интервал 5"), IIM4047 и IIM4978 в физических положениях 405220-34086738 хромосомы 9 (в данном документе "интервал 6"), IIM19 и IIM818 в физических положениях 1285447-29536061 хромосомы 10 (в данном документе "интервал 7") и их любых комбинаций (см. таблицы 1-7, показывающих SNP в пределах указанных хромосомных интервалов, которые ассоциированы с повышенной засухоустойчивостью. Положения аллелей заключенные в '***', а также выделенные жирным шрифтом и подчеркиванием, указывают на "аллели урожайности", которые расположены в пределах или в непосредственной близости от причинного гена в отношении засухоустойчивости и/или повышенной урожайности).
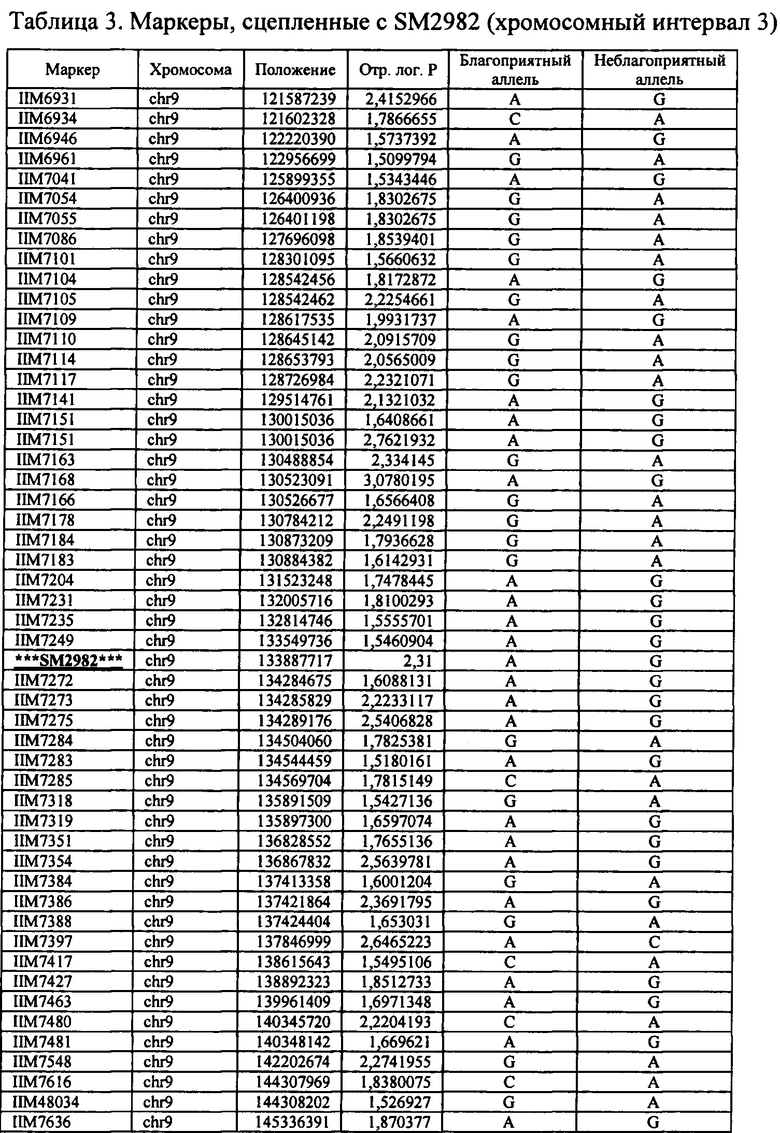
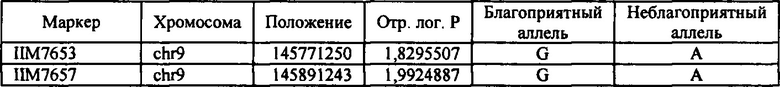
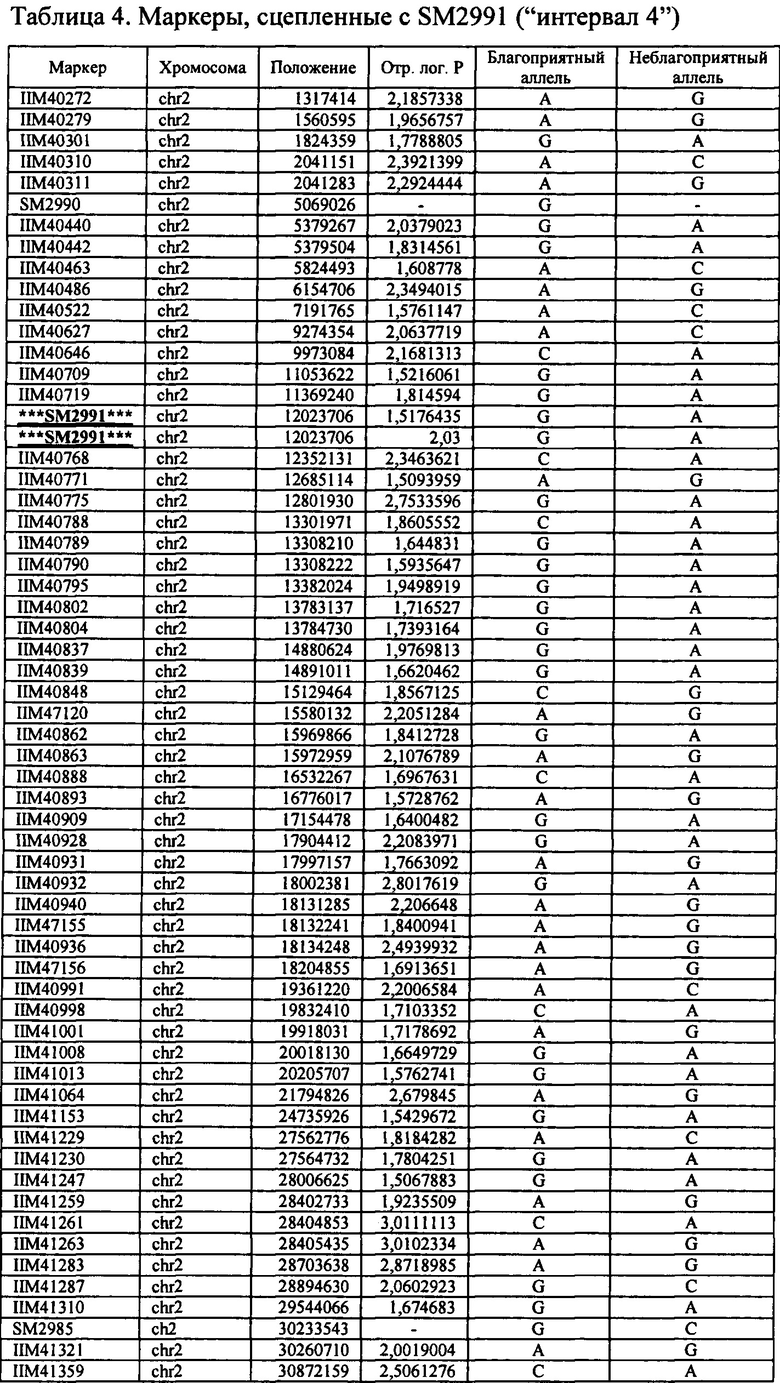
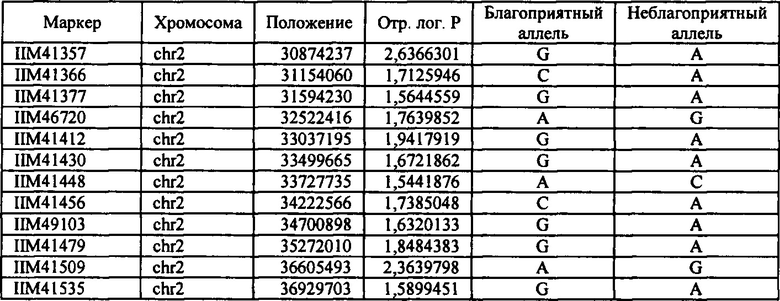
В некоторых вариантах осуществления предусмотрены способы получения засухоустойчивого растения маиса. Такие способы могут предусматривать выявление в идиоплазме маиса или в растении маиса присутствие маркера, ассоциированного с повышенной засухоустойчивостью (например, маркера в пределах любого хромосомного интервала или их комбинации, содержащих по меньшей мере один хромосомный интервал 1-15, определяемый в данном документе, любого маркера или их комбинации из маркеров, перечисленных в таблицах 1-7, или любого из аллелей урожайности 1-8 или маркеров, тесно ассоциированных с аллелями урожайности 1-8), и получение растения-потомка из указанных идиоплазмы или растения маиса, где указанное растение-потомок содержит указанный маркер, ассоциированный с повышенной засухоустойчивостью, и указанное растение-потомок также демонстрирует повышенную засухоустойчивость по сравнению с контрольным растением, не содержащим указанный маркер. В настоящем изобретении также предусмотрено семя, полученное от указанного растения-потомка.
В некоторых вариантах осуществления предусмотрено семя маиса, полученное за счет двух родительских линий маиса, где по меньшей мере одна родительская линия была идентифицирована или отобрана по признаку повышенной урожайности в условиях стресса, вызванного засухой, или повышенной урожайности в условиях, отличных от засухи, и также где урожайность представляет собой увеличение количества бушелей кукурузы на акр по сравнению с контрольным растением, и где по меньшей мере одна родительская линия была отобрана в соответствии со способом, включающим стадии: (а) выделения нуклеиновой кислоты из части растения маиса родительской линии; (b) выявления в нуклеиновой кислоте из (а) молекулярного маркера, который ассоциирован с засухоустойчивостью и/или повышенной урожайностью, где молекулярный маркер является тесно ассоциированным с любым из "аллелей урожайности 1-8", где тесно ассоциированный означает, что маркер находится в пределах 50 сМ, 40 сМ, 30 сМ, 20 сМ, 15 сМ, 10 сМ, 9 сМ, 8 сМ, 7 сМ, 6 сМ, 5 сМ, 4 сМ, 3 сМ, 2 сМ, 1 сМ или 0,5 сМ от указанного аллеля урожайности; и (с) отбора или идентификации растения маиса на основании присутствия указанного маркера на (b). В некоторых аспектах варианта осуществления молекулярный маркер из (b) находится в пределах хромосомного интервала, выбранного из любого из хромосомных интервалов 1-15, определяемых в данном документе.
В некоторых вариантах осуществления присутствие маркера, ассоциированного с повышенной засухоустойчивостью, выявляют с применением маркерного зонда. В некоторых таких вариантах осуществления присутствие маркера, ассоциированного с повышенной засухоустойчивостью, выявляют в продукте амплификации, полученном на основе образца нуклеиновой кислоты, выделенного из растения или идиоплазмы маиса. В некоторых вариантах осуществления маркер представляет собой гаплотип, и несколько зондов применяют для выявления аллелей, которые составляют гаплотип. В некоторых таких вариантах осуществления аллели, которые составляют гаплотип, выявляют в нескольких продуктах амплификации, полученных на основе образца нуклеиновой кислоты, выделенного из растения или идиоплазмы маиса.
В некоторых вариантах осуществления предусмотрены способы отбора засухоустойчивого растения или идиоплазмы маиса. Такие способы могут предусматривать скрещивание первого растения или идиоплазмы маиса со вторым растением или идиоплазмой маиса, где первое растение или идиоплазма маиса содержат маркер, ассоциированный с повышенной засухоустойчивостью, и отбор растения-потомка или идиоплазмы, которые имеют маркер (например, маркер, расположенный на расстоянии 50 сМ, 20 сМ, 10 сМ, 5 сМ, 2 сМ или 1 сМ от любого из хромосомных интервалов 1-15, маркер, расположенный в пределах хромосомного интервала или их комбинации, содержащих по меньшей мере один интервал 1-15, определяемый в данном документе, или любой маркер или их комбинацию из маркеров, перечисленных в таблицах 1-7, или аллели урожайности 1-8), который, как было продемонстрировано, ассоциирован с повышенной засухоустойчивостью и/или урожайностью.
В некоторых вариантах осуществления предусмотрены способы интрогрессии аллеля, ассоциированного с повышенной засухоустойчивостью, в растение маиса или идиоплазму маиса. Такие способы могут предусматривать скрещивание первого растения или идиоплазмы маиса, содержащих аллель, ассоциированный с повышенной засухоустойчивостью (например, любой аллель, идентифицированный в таблицах 1-7), с вторым растением или идиоплазмой маиса, у которых отсутствует указанный аллель, и повторное возвратное скрещивание растений-потомков, содержащих указанный аллель, со вторым растением или идиоплазмой маиса для получения засухоустойчивого растения или идиоплазмы маиса, содержащих аллель, ассоциированный с повышенной засухоустойчивостью. Потомков, содержащих аллель, ассоциированный с повышенной засухоустойчивостью, можно идентифицировать путем выявления в их геномах присутствия маркера, ассоциированного с указанным аллелем; например, маркера, расположенного в пределах хромосомного интервала (например, любого из хромосомных интервалов 1-15, или их части) или в пределах 50 сМ, 20 сМ, 10 сМ или меньше от аллелей урожайности 1-8, или их комбинации, содержащих по меньшей мере один хромосомный интервал 1-15, определяемый в данном документе, или любого маркера или их комбинации из маркеров, перечисленных в таблицах 1-7.
Также предусмотрены растения и/или идиоплазмы, идентифицированные, полученные или отобранные с помощью любого из способов по настоящему изобретению, также как любые потомки или семена, происходящие от растения или идиоплазмы, идентифицированных, полученных или отобранных с помощью этих способов, описанных в данном документе.
Также предусмотрены не встречающиеся в природе растения и/или идиоплазмы маиса с интрогрессированным (например, за счет селекции растений, трансгенной экспрессии или редактирования генома) в их геном любым из хромосомных интервалов 1-15, содержащим один или несколько маркеров, ассоциированных с повышенной засухоустойчивостью. В некоторых вариантах осуществления не встречающееся в природе растение и/или идиоплазма маиса представляют собой растение-потомок растения маиса, которое было отобрано для целей селекции на основании присутствия маркера, который ассоциирован с повышенной засухоустойчивостью и/или повышенной урожайностью в условиях достаточного наличия воды, и где указанный маркер расположен в пределах хромосомного интервала, который соответствует любому одному или нескольким из хромосомным интервалов 1, 2, 3, 4, 5, 6, 7 или их частям. В других вариантах осуществления не встречающееся в природе растение создают путем редактирования в пределах генома растения аллельного изменения, которое соответствует любому из аллелей урожайности 1-8 или благоприятных аллелей, идентифицированных в любой из таблиц 1-7, где аллельное изменение приводит к тому, что растение характеризуется повышенной засухоустойчивостью и/или повышенной урожайностью по сравнению с контрольным растением.
Также предусмотрены способы использования маркеров, ассоциированных с повышенной засухоустойчивостью. Такие маркеры могут содержать нуклеотидную последовательность, характеризующуюся по меньшей мере 85%, 90%, 95% или 99% идентичностью последовательности с любой из SEQ ID NO: 1-8, 17-66; их обратной комплементарной последовательности или их информативного или функционального фрагмента.
Также предусмотрены композиции, содержащие пару праймеров, которые способны амплифицировать образец нуклеиновой кислоты, выделенный из растения или идиоплазмы маиса, с получением маркера, ассоциированного с повышенной засухоустойчивостью. Такие композиции могут содержать, состоять фактически или состоять из одной из пар праймеров для амплификации, идентифицированных в таблице 8.
Маркер, ассоциированный с повышенной засухоустойчивостью, может содержать, состоять фактически и/или состоять из одного аллеля или комбинации аллелей в одном или нескольких генетических локусах (например, генетическом локусе, содержащем любую из SEQ ID NO: 1-8, 17-65 и/или аллели урожайности 1-8, определяемые в данном документе).
Другой вариант осуществления настоящего изобретения представляет собой способ отбора или идентификации растения маиса, характеризующегося повышенной засухоустойчивостью по сравнению с контрольным растением, где повышенная засухоустойчивость представляет собой повышенную урожайность в бушелях на акр по сравнению с контрольным растением, причем способ предусматривает стадии: а) выделения нуклеиновой кислоты из растения маиса; b) выявление в нуклеиновой кислоте из а) молекулярного маркера, который является тесно сцепленным и ассоциирован с засухоустойчивостью (например, любого маркера из таблиц 1-7); и с) идентификации или отбора линии маиса, характеризующейся повышенной засухоустойчивостью по сравнению с контрольным растением, на основании молекулярного маркера, выявленного на b). В некоторых вариантах осуществления маркер, выявленный на b), находится в пределах хромосомного интервала, выбранного из любого из хромосомных интервалов 1-15, определяемых в данном документе. В другом варианте осуществления маркер, выявленный на b), содержит любую из SEQ ID No: 17-24, где последовательность содержит любой благоприятный аллель, описываемый в таблицах 1-7. Дополнительные варианты осуществления включают хромосомный интервал, где любая из пар праймеров в таблице 8 отжигается с указанным интервалом, и ПЦР-амплификация приводит к образованию ампликона, позволяющего определить ассоциацию рассматриваемого маркера с повышенной засухоустойчивостью.
В другом варианте осуществления гены, хромосомные интервалы, маркеры и генетические локусы по настоящему изобретению можно комбинировать с маркерами, описанными в заявке на патент США 2011-0191892, включенной в данный документе посредством ссылки во всей своей полноте. Например, генетические локусы, содержащие любую из SEQ ID NO: 1-8; 17-77 или аллели, содержащиеся в них, которые ассоциированы с повышенной засухоустойчивостью и/или повышенной урожайностью маиса в условиях достаточного наличия воды, могут быть комбинированы с любым одним или несколькими из гаплотипов А-М, где гаплотипы А-М определены следующим образом:
i. гаплотип А содержит нуклеотид G в положении, которое соответствует положению 115 из SEQ ID NO: 65, нуклеотид А в положении, которое соответствует положению 270 из SEQ ID NO: 65, нуклеотид Т в положении, которое соответствует положению 301 из SEQ ID NO: 65, и нуклеотид А в положении, которое соответствует положению 483 из SEQ ID NO: 1, на хромосоме 8 в геноме первого растения;
ii. гаплотип В содержит делецию в положениях 4497-4498 из SEQ ID NO: 66, нуклеотид G в положении, которое соответствует положению 4505 из SEQ ID NO: 66, нуклеотид Т в положении, которое соответствует положению 4609 из SEQ ID NO: 66, нуклеотид А в положении, которое соответствует положению 4641 из SEQ ID NO: 66, нуклеотид Т в положении, которое соответствует положению 4792 из SEQ ID NO: 66, нуклеотид Т в положении, которое соответствует положению 4836 из SEQ ID NO: 66, нуклеотид С в положении, которое соответствует положению 4844 из SEQ ID NO: 66, нуклеотид G в положении, которое соответствует положению 4969 из SEQ ID NO: 66, и тринуклеотид ТСС в положении, которое соответствует положениям 4979-4981 из SEQ ID NO: 66, на хромосоме 8 в геноме первого растения;
iii. гаплотип С содержит нуклеотид А в положении, которое соответствует положению 217 из SEQ ID NO: 67, нуклеотид G в положении, которое соответствует положению 390 из SEQ ID NO: 67, и нуклеотид А в положении, которое соответствует положению 477 из SEQ ID NO: 67, на хромосоме 2 в геноме первого растения;
iv. гаплотип D содержит нуклеотид G в положении, которое соответствует положению 182 из SEQ ID NO: 68, нуклеотид А в положении, которое соответствует положению 309 из SEQ ID NO: 68, нуклеотид G в положении, которое соответствует положению 330 из SEQ ID NO: 68, и нуклеотид G в положении, которое соответствует положению 463 из SEQ ID NO: 68, на хромосоме 8 в геноме первого растения;
v. гаплотип Е содержит нуклеотид С в положении, которое соответствует положению 61 из SEQ ID NO: 69, нуклеотид С в положении, которое соответствует положению 200 из SEQ ID NO: 69, и делецию из девяти нуклеотидов в положениях, которые соответствуют положениям 316-324 из SEQ ID NO: 69, на хромосоме 5 в геноме первого растения;
vi. гаплотип F содержит нуклеотид G в положении, которое соответствует положению 64 из SEQ ID NO: 70, и нуклеотид Т в положении, которое соответствует положению 254 из SEQ ID NO: 70, на хромосоме 8 в геноме первого растения;
vii. гаплотип G содержит нуклеотид С в положении, которое соответствует положению 98 из SEQ ID NO: 71, нуклеотид Т в положении, которое соответствует положению 147 из SEQ ID NO: 71, нуклеотид С в положении, которое соответствует положению 224 из SEQ ID NO: 71, и нуклеотид Т в положении, которое соответствует положению 496 из SEQ ID NO: 71, на хромосоме 9 в геноме первого растения;
viii. гаплотип Н содержит нуклеотид Т в положении, которое соответствует положению 259 из SEQ ID NO: 72, нуклеотид Т в положении, которое соответствует положению 306 из SEQ ID NO: 72, нуклеотид А в положении, которое соответствует положению 398 из SEQ ID NO: 72, и нуклеотид С в положении, которое соответствует положению 1057 из SEQ ID NO: 72, на хромосоме 4 в геноме первого растения;
ix. гаплотип I содержит нуклеотид С в положении, которое соответствует положению 500 из SEQ ID NO: 73, нуклеотид G в положении, которое соответствует положению 568 из SEQ ID NO: 73, и нуклеотид Т в положении, которое соответствует положению 698 из SEQ ID NO: 73, на хромосоме 6 в геноме первого растения;
x. гаплотип J содержит нуклеотид А в положении, которое соответствует положению 238 из SEQ ID NO: 74, делецию из нуклеотидов, которые соответствуют положениям 266-268 из SEQ ID NO: 74, и нуклеотид С в положении, которое соответствует положению 808 из SEQ ID NO: 74, в геноме первого растения;
xi. гаплотип K содержит нуклеотид С в положении, которое соответствует положению 166 из SEQ ID NO: 75, и нуклеотид А в положении, которое соответствует положению 224 из SEQ ID NO: 75, нуклеотид G в положении, которое соответствует положению 650 из SEQ ID NO: 75, и нуклеотид G в положении, которое соответствует положению 892 из SEQ ID NO: 75, на хромосоме 8 в геноме первого растения;
xii. гаплотип L содержит нуклеотид С в положениях, которые соответствуют положениям 83, 428, 491 и 548 из SEQ ID NO: 76, на хромосоме 9 в геноме первого растения; и
xiii. гаплотип М содержит нуклеотид С в положении, которое соответствует положению 83 в SEQ ID NO: 77, нуклеотид А в положении, которое соответствует положению 119 из SEQ ID NO: 77, и нуклеотид Т в положении, которое соответствует положению 601 из SEQ ID NO: 77.
Таким образом, в некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, предусмотрен способ пакетирования гаплотипов, выбранных из группы, состоящей из любого из гаплотипов А, В, С, D, Е, F, G, Н, I, J, K, L и М с маркером, выбранным из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 и SM2984 и тесно ассоциированных с ними маркеров, таких как маркеры в таблицах 1-7; или маркеров, тесно сцепленных с SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 и SM2984, или маркеров, содержащих любую из SEQ ID No: 17-24. Дополнительно предусмотрены растения маиса, содержащие в своем геноме пакеты из гаплотипов и/или локусов, которые не встречаются в природе, где пакеты содержат любой из определяемых гаплотипов А-М в комбинации с любым из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 и SM2984. В некоторых случаях растения маиса, содержащие эти уникальные пакеты, которые не встречаются в природе (например, содержащие комбинацию из гаплотипов А-М или локусов SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 и SM2984), представляют собой гибридные растения маиса, и в некоторых случаях гибридное растение маиса содержит в своем геноме активный трансген для обеспечения одного из устойчивости к гербицидам и/или устойчивости к насекомым.
Таким образом, в некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, предусмотрены способы получения гибридного растения с повышенной засухоустойчивостью. В некоторых вариантах осуществления способ предусматривает (а) обеспечение первого растения, содержащего первый генотип, содержащий любой из гаплотипов А-М: (b) обеспечение второго растения, содержащего второй генотип, содержащий любой из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 и SM2984, где второе растение содержит по меньшей мере один маркер из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 и SM2984, который не присутствует в первом растении; (с) скрещивание первого растения и второго растения маиса с получением поколения F1; идентификацию одного или нескольких представителей поколения F1, которые содержат требуемый генотип, содержащий любую комбинацию из гаплотипов А-М и любых маркеров из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982, и SM2984, где требуемый генотип отличается как от первого генотипа из (а), так и от второго генотипа из (b), при этом получают гибридное растение с повышенной засухоустойчивостью. В некоторых аспектах варианта осуществления гибридное растение из (b) дополнительно содержит в пределах своего генома трансген для обеспечения устойчивости к гербицидам и/или устойчивости к насекомым. В некоторых аспектах гибридное растение из (b) представляет собой элитную линию маиса.
В другом варианте осуществления объектов, раскрытых в настоящем изобретении, раскрыт способ получения растения маиса, характеризующегося повышенной засухоустойчивостью по сравнению с контрольным растением, где урожайность представляет собой увеличение количества бушелей на акр (в некоторых вариантах осуществления YGSMN), причем способ предусматривает стадии: а) выделения нуклеиновой кислоты из первого растения маиса; b) выявления в нуклеиновой кислоте из а) молекулярного маркера, ассоциированного с повышенной засухоустойчивостью (например, любого из маркеров, описанных в таблицах 1-7, или тесно ассоциированных маркеров), где маркер расположен в пределах хромосомного интервала 1-15; или где хромосомный интервал определяется как 50 сМ, 40 сМ, 30 сМ, 20 сМ, 10 сМ, 9 сМ, 8 сМ, 7 сМ, 6 сМ, 5 сМ, 4 сМ, 3 сМ, 2 сМ, 1 сМ или 0,5 сМ или меньше от любого из аллелей урожайности 1-8; или хромосомный интервал содержит любую из SEQ ID No 17-24; или маркер является тесно ассоциированным с соответствующим маркером, описанным в таблицах 1-7; с) отбора первого растения маиса на основании маркера, выявленного на b); d) скрещивания первого растения маиса со вторым растением маиса, не содержащим маркер из b); е) получения растения-потомка, полученного на основе скрещивания на d), где растение-потомок имеет интрогрессированный в его геном маркер из b), с получением тем самым растения маиса, характеризующегося повышенной засухоустойчивостью по сравнению с контрольным растением. В некоторых аспектах семя получено за счет варианта осуществления, где семя содержит в своем геноме маркер из b).
В другом варианте осуществления объектов, раскрытых в настоящем изобретении, раскрыт способ получения растения, характеризующегося повышенной засухоустойчивостью, повышенной урожайностью в условиях засухи или повышенной урожайностью в условиях, отличных от засухи, по сравнению с контрольным растением, причем способ предусматривает стадии: а) в растительной клетке редактирования генома растения (т.е. за счет CRISPR, TALEN или мегануклеаз) с тем, чтобы он содержал молекулярный маркер (например, SNP), ассоциированный с повышенной засухоустойчивостью, повышенной урожайностью в условиях засухи или повышенной урожайностью в условиях, отличных от засухи, где молекулярный маркер представляет собой любой маркер (например, благоприятный аллель), описываемый в таблицах 1-7, и дополнительно, где геном растения не имел указанный молекулярный маркер ранее; b) получения растения или растительного каллюса из растительной клетки из а). В частности, редактирование предусматривает любой из аллелей урожайности 1-8 или их тесно ассоциированных аллелей. В другом аспекте варианта осуществления редактирование предполагает получение гена, характеризующегося 70%, 80%, 85%, 90%, 92%, 95%, 98%, 99% или 100% гомологией последовательности или идентичностью последовательности с геном, содержащим SEQ ID No: 1-8.
В некоторых вариантах осуществления гибридное растение с повышенной засухоустойчивостью содержит каждый из гаплотипов А-М, которые присутствуют в первом растении, а также по меньшей мере один дополнительный локус, выбранный из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 и SM2984 (или маркер в пределах любого из хромосомных интервалов 1-15, который ассоциирован с одной из повышенной засухоустойчивости и/или повышенной урожайности в условиях достаточного наличия воды, где урожайность представляет собой увеличение количества бушелей на акр, или маркер, содержащий SEQ ID No 17-24), который присутствует во втором растении. В некоторых вариантах осуществления первое растение представляет собой рекуррентного родителя, содержащего по меньшей мере один из гаплотипов А-М, а второе растение представляет собой донора, который содержит по меньшей мере один маркер из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 или SM2984, который не присутствует в первом растении. В некоторых вариантах осуществления первое растение является гомозиготным по меньшей мере по двум, трем, четырем или пяти из гаплотипов А-М. В некоторых вариантах осуществления гибридное растение содержит по меньшей мере три, четыре, пять, шесть, семь, восемь или девять из гаплотипов А-М и маркеры из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 или SM2984 или любого из аллелей урожайности 1-8.
В некоторых вариантах осуществления можно идентифицировать засухоустойчивое растение маиса путем генотипирования одного или нескольких представителей поколения F1, полученного за счет скрещивания первого растения и второго растения, в отношении каждого из гаплотипов А-М и маркеров из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 и SM2984, присутствующих либо в первом растении, либо во втором растении. В некоторых вариантах осуществления первое растение и второе растение являются растениями Zea mays, и в других случаях первое и второе растение являются инбредными растениями Zea mays.
В некоторых вариантах осуществления "повышенная оптимизация потребления воды" обеспечивает повышенную или стабилизированную урожайность в обстановке недостатка воды по сравнению с контрольным растением. Растения маиса, характеризующиеся улучшением оптимизации потребления воды, можно отбирать, идентифицировать или получать с применением любого из маркеров, перечисленных в таблицах 1-7, или маркера в пределах хромосомных интервалов 1-15. В некоторых вариантах осуществления гибрид с повышенной оптимизацией потребления воды можно высаживать при большей густоте посева. В некоторых вариантах осуществления гибрид с повышенной оптимизацией потребления воды обеспечивает отсутствие груза для урожая, когда он находится при благоприятных уровнях обеспеченности влагой. В еще одном варианте осуществления растения, содержащие любой из маркеров или хромосомных интервалов, идентифицированных в таблицах 1-7, могут обеспечивать что-либо из повышенной засухоустойчивости или повышенной урожайности по сравнению с контрольным растением или дополнительную повышенную урожайность в условиях, отличных от засухи, или условиях достаточного наличия воды, где урожайность представляет собой увеличение количества бушелей кукурузы на акр (т.е. YGSMN).
В некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, также предусмотрены гибридные растения Zea mays, полученные с помощью способов, раскрытых в данном документе, или их клетку, культуру тканей, семя или часть растения.
В некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, также предусмотрены инбредные растения Zea mays, полученные с помощью возвратного скрещивания, и/или самоопыления, и/или получения двойных гаплоидов из гибридных растений Zea mays, раскрытых в данном документе, или их клетка, культура тканей, семя или часть.
В некоторых вариантах осуществления растения маиса, характеризующиеся повышенной засухоустойчивостью, идентифицируют путем генотипирования одного или нескольких представителей поколения F1, полученного за счет скрещивания первого растения и второго растения, в отношении каждого из хромосомных интервалов, маркеров и/или их комбинации, показанных в таблицах 1-7 или содержащихся в любой из или комбинации из SEQ ID NO: 1-8; 17-65, присутствующих либо в первом растении, либо во втором растении. В некоторых вариантах осуществления первое растение и второе растение представляют собой растения Zea mays. В других вариантах осуществления первое растение или второе растение представляют собой одно из инбредной линии Zea mays, или гибридной линии Zea mays, или элитной линии Zea mays.
В некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, также предусмотрены гибридные или инбредные растения Zea mays, которые были модифицированы для включения трансгена. В некоторых вариантах осуществления трансген кодирует продукт гена, который придает устойчивость к гербициду, выбранному из глифосата, сульфонилмочевины, имидазолинона, дикамбы, глюфосината, феноксипропионовой кислоты, циклошексома, триазина, бензонитрила и броксинила. Например, любое гибридное или инбредное растение Zea mays содержит в своем геноме трансген, кодирующий любой из трансгенов устойчивости к глифосату, сульфонилмочевине, имидазолинону, дикамбе, глюфосинату, феноксипропионовой кислоте, циклошексому, триазину, бензонитрилу и броксинилу, и где в геном указанного растения были введены с помощью селекции растений, трансгенной экспрессии или редактирования генома любая из SEQ ID No 1-8 или любой из аллелей урожайности 1-8.
В некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, также предусмотрены способы идентификации растений Zea mays, содержащих по меньшей мере один аллель, ассоциированный с повышенной засухоустойчивостью, раскрытый в данном документе (например, любой маркер, тесно ассоциированный с аллелями, описанными в таблицах 1-7). В некоторых вариантах осуществления способы предусматривают (а) генотипирование и идентификацию по меньшей мере одного растения Zea mays по меньшей мере с одним маркером на основе нуклеиновой кислоты, содержащим любую из SEQ ID NO: 1-8; 17-60; и (b) отбор по меньшей мере одного растения Zea mays, содержащего аллель, ассоциированный с засухоустойчивостью, идентифицированный на b).
В некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, также предусмотрены растения Zea mays, полученные путем интрогрессии представляющего интерес аллеля из локуса, ассоциированный с повышенной засухоустойчивостью, в идиоплазму Zea mays. В некоторых вариантах осуществления интрогрессия предусматривает (а) отбор растения Zea mays, которое содержит представляющий интерес аллель из локуса, ассоциированного с повышенной засухоустойчивостью, где локус, ассоциированный с повышенной засухоустойчивостью, содержит нуклеотидную последовательность, которая по меньшей мере на 80%, 85%, 90%, 95%, 98% или 100% идентична любой из SEQ ID NO: 1-8; 17-60, или где нуклеотидная последовательность содержит любой из аллелей урожайности 1-7 или их комбинацию; и (b) интрогрессию представляющего интерес аллеля в идиоплазму Zea mays, в которой отсутствует данный аллель.
В другом варианте осуществления в настоящем изобретении предусмотрена идиоплазма маиса, которая была подвергнута обогащению любым из хромосомных интервалов 1-15 или аллелей урожайности 1-7, где обогащение предусматривает стадии идентификации или отбора линий с указанными хромосомными интервалами или аллелями урожайности, и скрещивания этих линий с линиями, у которых отсутствуют указанные интервалы или их части, и возвратного скрещивания для создания инбредных линий с указанными интервалами или аллелями урожайности, а затем использования указанных инбредных линий в системе селекции растений для создания коммерческой популяции маиса, обогащенной в отношении указанного интервала или его аллелей урожайности (например, коммерческой популяции гибридного маиса, в которой больше 30%, 40% или более 50% гибридов, обогащены с помощью указанного интервала или аллелей урожайности по сравнению с 5-летней исторической родословной указанной популяции гибридного маиса, характеризующего <30% обогащением по указанному интервалу или аллелям урожайности.
В некоторых вариантах осуществления подразумевается способ идентификации и/или отбора растения или части растения маиса, характеризующихся повышенной урожайностью в условиях, отличных от засухи, повышенной стабильностью урожайности в условиях засухи и/или повышенной засухоустойчивостью, который предусматривает: выявление в растении или части растения маиса аллеля по меньшей мере одного маркерного локуса, который ассоциирован с повышенной урожайностью в условиях, отличных от засухи, повышенной стабильностью урожайности в условиях засухи и/или повышенной засухоустойчивостью растения, где указанный по меньшей мере один маркерный локус расположен в пределах хромосомного интервала, выбранного из группы, состоящей из:
(a) хромосомного интервала на хромосоме 1 маиса, определяемого от положения пары оснований (п. о.) 272937470 до положения пары оснований (п. о.) 272938270 включительно (в данном документе "интервал 8");
(b) хромосомного интервала на хромосоме 2 маиса, определяемого от положения пары оснований (п. о.) 12023306 до положения пары оснований (п. о.) 12024104 включительно (в данном документе "интервал 9");
(c) хромосомного интервала на хромосоме 3 маиса, определяемого от положения пары оснований (п. о.) 225037202 до положения пары оснований (п. о.) 225038002 включительно (в данном документе "интервал 10");
(d) хромосомного интервала на хромосоме 3 маиса, определяемого от положения пары оснований (п. о.) 225340531 до положения пары оснований (п. о.) 225341331 (в данном документе "интервал 11");
(e) хромосомного интервала на хромосоме 5 маиса, определяемого от положения пары оснований (п. о.) 159120801 до положения пары оснований (п. о.) 159121601 включительно (в данном документе "интервал 12");
(f) хромосомного интервала на хромосоме 9 маиса, определяемого от положения пары оснований (п. о.) 12104536 до положения пары оснований (п. о.) 12105336 включительно (в данном документе "интервал 13"); (g) хромосомного интервала на хромосоме 9 маиса, определяемого от положения пары оснований (п. о.) 225343590 до положения пары оснований (п. о.) 225340433 включительно (в данном документе "интервал 14");
(е) хромосомного интервала на хромосоме 10 маиса, определяемого от положения пары оснований (п. о.) 14764415 до положения пары оснований (п. о.) 14765098 включительно (в данном документе "интервал 15"). В предпочтительном варианте осуществления хромосомные интервалы 8-14 дополнительно содержат соответствующий аллель урожайности 1-7, определяемый в данном документе.
В дополнительных вариантах осуществления способ идентификации и/или отбора растения или части растения маиса, характеризующихся повышенной урожайностью в условиях, отличных от засухи, повышенной стабильностью урожайности в условиях засухи и/или повышенной засухоустойчивостью, предусматривает: выявление в растении или части растения маиса аллеля по меньшей мере одного маркерного локуса, который ассоциирован с повышенной урожайностью в условиях, отличных от засухи, повышенной стабильностью урожайности в условиях засухи и/или повышенной засухоустойчивостью растения, где указанный по меньшей мере один маркер выбран из группы следующих причинных аллелей, или маркер расположен в пределах 50 сМ, 40 сМ, 30 сМ, 20 сМ, 15 сМ, 10 сМ, 9 сМ, 8 сМ, 7 сМ, 6 сМ, 5 сМ, 4 сМ, 3 сМ, 2 сМ, 1 сМ или 0,5 сМ от них:
положения п. о. 272937870 на хромосоме 1, содержащего аллель G;
положения п. о. 12023706 на хромосоме 2, содержащего аллель G;
положения п. о. 225037602 на хромосоме 3, содержащего аллель А;
положения п. о. 225340931 на хромосоме 3, содержащего аллель А;
положения п. о. 159121201 на хромосоме 5, содержащего аллель G;
положения п. о. 12104936 на хромосоме 9, содержащего аллель С;
положения п. о. 133887717 на хромосоме 9, содержащего аллель А; и
положения п. о. 4987333 на хромосоме 10, содержащего аллель G; или любой их комбинации.
В другом варианте осуществления предусмотрен способ отбора засухоустойчивого растения маиса, причем способ предусматривает стадии: а) выделения нуклеиновой кислоты из растительной клетки; b) выявления в указанной нуклеиновой кислоте молекулярного маркера, ассоциированного с повышенной засухоустойчивостью, где указанный маркер находится в пределах хромосомного интервала, содержащего любой из хромосомных интервалов 1-15, определяемых в данном документе; и с) отбора или идентификации растения маиса, характеризующегося повышенной засухоустойчивостью, на основе выявления маркера на b). В некоторых дополнительные варианты осуществления при этом соответствующий хромосомный интервал содержит любой из следующих аллей:
положение п. о. 272937870 на хромосоме 1, содержащее аллель G;
положение п. о. 12023706 на хромосоме 2, содержащее аллель G;
положение п. о. 225037602 на хромосоме 3, содержащее аллель А;
положение п. о. 225340931 на хромосоме 3, содержащее аллель А;
положение п. о. 159121201 на хромосоме 5, содержащее аллель G;
положение п. о. 12104936 на хромосоме 9, содержащее аллель С;
положение п. о. 133887717 на хромосоме 9, содержащее аллель А; и
положение п. о. 4987333 на хромосоме 10, содержащее аллель G;
любой аллель, перечисленный в таблицах 1-7; или любую их комбинацию.
В некоторых вариантах осуществления в настоящем изобретении предусмотрены способы получения гибридного растения маиса с повышенной урожайностью, где урожайность повышена либо в условиях засухи, либо в условиях, отличных от засухи, и повышенная урожайность представляет собой увеличение количества бушелей на акр кукурузы по сравнению с контролем, при этом способ предусматривает стадии: (а) идентификации первого растения маиса, содержащего первый генотип, путем идентификации любого из маркеров SM2987, SM2996, SM2982, SM2991, SM2995, SM2973, SM2980 или SM2984, аллелей урожайности 1-8 или любых их тесно ассоциированных маркеров (например, любых маркеров в таблицах 1-7); (b) идентификации второго растения маиса, содержащего второй генотип, путем идентификации любого из маркеров SM2987, SM2996, SM2982, SM2991, SM2995, SM2973, SM2980 или SM2984, или аллелей урожайности 1-8, не содержащихся в первом растении маиса, с) скрещивания первого растения маиса и второго растения маиса с получением поколения F1; и (d) отбора одного или нескольких представителей поколения F1, которое содержит требуемый генотип, содержащий любую комбинацию маркеров SM2987, SM2996, SM2982, SM2991, SM2995, SM2973, SM2980 или SM2984, где требуемый генотип отличается как от первого генотипа из (а), так и от второго генотипа из (b), при этом получают гибридное растение маиса с повышенной урожайностью в бушелях на акр.
В одном варианте осуществления настоящего изобретения предусмотрено неприродное гибридное растение, содержащее молекулу нуклеиновой кислоты, выбранную из группы, состоящей из SEQ ID NO: 17-24 или их фрагментов, аллелей урожайности 1-8 или комплементарных им последовательностей.
В настоящем изобретении также предусмотрено растение, содержащее аллели SM2987, SM2996, SM2982, SM2991, SM2995, SM2973, SM2980 или SM2984, или их фрагменты и комплементарные последовательности, а также любое растение, содержащее любую комбинацию одного или нескольких локусов засухоустойчивости, выбранного из группы, состоящей из SEQ ID NO: 17-24, где указанные локусы засухоустойчивости ассоциированы с повышенной засухоустойчивостью. Такие аллели могут быть гомозиготными или гетерозиготными.
В другом варианте осуществления настоящего изобретения предусмотрены способы введения в геном растения гена, который обеспечивает повышенную засухоустойчивость или повышенную урожайность указанного растения. Подразумевается, что гены можно вводить посредством традиционных способов селекции растений, трансгенной экспрессии, путем введения мутации, как, например, с помощью этилметансульфоната (ESM), или за счет подходов редактирования генов, таких как TALEN, CRISPR, мегануклеаза и т.д. Не ограничиваясь какой-либо теорией, в некоторых вариантах осуществления нуклеотидная последовательность содержит любую одну или несколько генных моделей, перечисленных в таблице 9 ниже, или SEQ ID No 1-8 можно вводить в геном растения для создания растений с повышенной урожайностью и/или повышенной засухоустойчивостью по сравнению с контрольным растением. Также подразумевается, что можно аналогично вводить причинный аллель, обуславливающий повышенную урожайность, где причинный аллель выбран из аллелей, перечисленных в любой из таблиц 1-7.

В одном варианте осуществления предусматриваются композиции и способы получения растений, характеризующихся повышенной засухоустойчивостью, которые можно получить с применением любого из молекулярных маркеров, описываемых в таблицах 1-7. Например, растение маиса можно идентифицировать, отбирать или получать за счет идентификации и/или отбора аллеля, который ассоциирован с повышенной засухоустойчивостью, как показано в таблицах 1-7.
В другом аспекте настоящего изобретения трансгенные растения, характеризующиеся повышенной устойчивостью к засухе и/или повышенной урожайностью, можно получать путем обеспечения функционального сцепления любого из генов в таблице 9, или SEQ ID No: 1-8, или их гомологов/ортологов, с растительным промотором (конститутивным или тканеспецифическим) и экспрессии указанного гена в растении. Например, подразумевается, что указанные гены можно экспрессировать с применением либо конститутивной, либо тканеспецифической/тканепредпочтительной экспрессии. Не ограничиваясь каким-либо примером, подразумевается, что можно будет нацеливать экспрессию, например, в початок, цветоножку, репродуктивную ткань, плод, семя или другие части растения кукурузы для получения трансгенных растений, характеризующихся повышенной урожайностью и/или засухоустойчивостью.
Эти и другие аспекты настоящего изобретения изложены более подробно в нижеследующем описании настоящего изобретения.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
ФИГ. 1 представляет собой столбчатую диаграмму, демонстрирующую, что трансгенные растения, экспрессирующие GRMZM2G027059 (конструкция 23294), содержат значительно большее количество общего хлорофилла по сравнению с контрольными (CK) растениями.
ФИГ. 2 представляет собой столбчатую диаграмму, демонстрирующую, что трансгенные растения с экспрессией GRMZM2G156365 Т демонстрируют повышенное содержание сахаров, вовлеченных в образование пектина (данные для трансформантов относительно возрастания по сравнению с контролями).
ФИГ. 3 представляет собой метаболический профиль трансгенных растений Т1, сверэкспрессирующих GRMZM2G094428 (колонки справа представляют собой контроли дикого типа: сверхэкспрессия данного гена у Arabidopsis снижает уровни двух главных субстратов для образования лигнина и повышает уровень рецептора сложных эфиров спермидина).
ФИГ. 4 представляет собой метаболический профиль транс генных растений Т1, сверхэкспрессирующих GRMZM2G416751 (контроли находятся справа; сверхэкспрессия данного гена у Arabidopsis снижает экспрессию глюкуроната, 3-дезоксиоктулозоната и синапата).
ФИГ. 5 представляет собой столбчатую диаграмму, демонстрирующую, что трансгенные растения, экспрессирующие GRMZM2G467169 (конструкция 23403), содержат значительно большее количество общего хлорофилла по сравнению с контрольными (CK) растениями.
ФИГ. 6 представляет собой столбчатую диаграмму, демонстрирующую, что трансгенные растения, экспрессирующие GRMZM5G862107 (конструкция 23292), характеризуются значительно более высокой экспрессией HsfA2 в трансформантах 2 по сравнению с контролями дикого типа, что указывает на возможную роль в переносимости теплового стресса.
КРАТКОЕ ОПИСАНИЕ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
В настоящее раскрытие включены несколько нуклеотидных и/или аминокислотных последовательностей. На протяжении раскрытия и сопутствующего перечня последовательностей для идентификации нуклеотидов используют стандарт WIPO ST.25 (1998; далее в данном документе "стандарт ST.25"). Стандарт идентификации нуклеотидов обобщается ниже:


Дополнительно, независимо от того, отмечено специально или нет, в случае каждого упоминания "n" в перечне последовательностей, понятно, что каждый отдельный "n" (включая несколько или все n в последовательности из идущих подряд n) может представлять собой а, с, g, t/u, неизвестный, или другой, или может отсутствовать. Так, если в перечне последовательностей специально не определено иное, в некоторых вариантах осуществления "n" может представлять собой отсутствие нуклеотида.
SEQ ID NO: 1 представляет собой нуклеотидную последовательность кДНК гена оптимизации потребления воды GRMZM2G027059, расположенного на хромосоме 1 Zm в пределах хромосомных интервалов 1 и 8.
SEQ ID NO: 2 представляет собой нуклеотидную последовательность кДНК гена оптимизации потребления воды GRMZM2G156366, расположенного на хромосоме 2 Zm в пределах хромосомных интервалов 4 и 9.
SEQ ID NO: 3 представляет собой нуклеотидную последовательность кДНК гена оптимизации потребления воды GRMZM2G134234, расположенного на хромосоме 3 Zm в пределах хромосомных интервалов 2 и 10.
SEQ ID NO: 4 представляет собой нуклеотидную последовательность кДНК гена оптимизации потребления воды GRMZM2G094428, расположенного на хромосоме 3 Zm в пределах хромосомных интервалов 2 и 11.
SEQ ID NO: 5 представляет собой нуклеотидную последовательность кДНК гена оптимизации потребления воды GRMZM2G416751, расположенного на хромосоме 5 Zm в пределах хромосомных интервалов 5 и 12.
SEQ ID NO: 6 представляет собой нуклеотидную последовательность кДНК гена оптимизации потребления воды GRMZM2G467169, расположенного на хромосоме 9 Zm в пределах хромосомных интервалов 6 и 13.
SEQ ID NO: 7 представляет собой нуклеотидную последовательность кДНК гена оптимизации потребления воды GRMZM5G862107, расположенного на хромосоме 9 Zm в пределах хромосомных интервалов 3 и 14.
SEQ ID NO: 8 представляет собой нуклеотидную последовательность кДНК гена оптимизации потребления воды GRMZM2G050774, расположенного на хромосоме 10 Zm в пределах хромосомных интервалов 7 и 15.
SEQ ID NO: 9 представляет собой последовательность белка, кодируемого геном оптимизации потребления воды GRMZM2G027059.
SEQ ID NO: 10 представляет собой последовательность белка, кодируемого геном оптимизации потребления воды GRMZM2G156365.
SEQ ID NO: 11 представляет собой последовательность белка, кодируемого геном оптимизации потребления воды GRMZM2G134234.
SEQ ID NO: 12 представляет собой последовательность белка, кодируемого геном оптимизации потребления воды GRMZM2G094428.
SEQ ID NO: 13 представляет собой последовательность белка, кодируемого геном оптимизации потребления воды GRMZM2G416751.
SEQ ID NO: 14 представляет собой последовательность белка, кодируемого геном оптимизации потребления воды GRMZM2G467169.
SEQ ID NO: 15 представляет собой последовательность белка, кодируемого геном оптимизации потребления воды GRMZM5G862107.
SEQ ID NO: 16 представляет собой последовательность белка, кодируемого геном оптимизации потребления воды GRMZM2G050774.
SEQ ID NO: 17 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды SM2987, подпоследовательности которого могут быть амплифицированы с хромосомы 1 генома Zea mays с применением полимеразной цепной реакции с праймерами для амплификации, изложенными в таблице 8.
SEQ ID NO: 18 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды SM2991, подпоследовательности которого могут быть амплифицированы с хромосомы 2 генома Zea mays с применением полимеразной цепной реакции с праймерами для амплификации, изложенными в таблице 8.
SEQ ID NO: 19 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды SM2995, подпоследовательности которого могут быть амплифицированы с хромосомы 3 генома Zea mays с применением полимеразной цепной реакции с праймерами для амплификации, изложенными в таблице 8.
SEQ ID NO: 20 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды SM2996, подпоследовательности которого могут быть амплифицированы с хромосомы 3 генома Zea mays с применением полимеразной цепной реакции с праймерами для амплификации, изложенными в таблице 8.
SEQ ID NO: 21 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды SM2973, подпоследовательности которого могут быть амплифицированы с хромосомы 5 генома Zea mays с применением полимеразной цепной реакции с праймерами для амплификации, изложенными в таблице 8.
SEQ ID NO: 22 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды SM2980, подпоследовательности которого могут быть амплифицированы с хромосомы 9 генома Zea mays с применением полимеразной цепной реакции с праймерами для амплификации, изложенными в таблице 8.
SEQ ID NO: 23 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды SM2982, подпоследовательности которого могут быть амплифицированы с хромосомы 9 генома Zea mays с применением полимеразной цепной реакции с праймерами для амплификации, изложенными в таблице 8.
SEQ ID NO: 24 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды SM2984, подпоследовательности которого могут быть амплифицированы с хромосомы 10 генома Zea mays с применением полимеразной цепной реакции с праймерами для амплификации, изложенными в таблице 8.
SEQ ID NO: 25 представляет собой праймер для амплификации SM2987.
SEQ ID NO: 26 представляет собой праймер для амплификации SM2987.
SEQ ID NO: 27 представляет собой зонд для SM2987.
SEQ ID NO: 28 представляет собой зонд для SM2987.
SEQ ID NO: 29 представляет собой праймер для амплификации SM2991.
SEQ ID NO: 30 представляет собой праймер для амплификации SM2991.
SEQ ID NO: 31 представляет собой зонд для SM2991.
SEQ ID NO: 32 представляет собой зонд для SM2991.
SEQ ID NO: 33 представляет собой праймер для амплификации SM2995.
SEQ ID NO: 34 представляет собой праймер для амплификации SM2995.
SEQ ID NO: 35 представляет собой зонд для SM2995.
SEQ ID NO: 36 представляет собой зонд для SM2995.
SEQ ID NO: 37 представляет собой праймер для амплификации SM2996.
SEQ ID NO: 38 представляет собой праймер для амплификации SM2996.
SEQ ID NO: 39 представляет собой зонд для SM2996.
SEQ ID NO: 40 представляет собой зонд для SM2996.
SEQ ID NO: 41 представляет собой праймер для амплификации SM2973.
SEQ ID NO: 42 представляет собой праймер для амплификации SM2973.
SEQ ID NO: 43 представляет собой зонд для SM2973.
SEQ ID NO: 44 представляет собой зонд для SM2973.
SEQ ID NO: 45 представляет собой праймер для амплификации SM2980.
SEQ ID NO: 46 представляет собой праймер для амплификации SM2980.
SEQ ID NO: 47 представляет собой зонд для SM2980.
SEQ ID NO: 48 представляет собой зонд для SM2980.
SEQ ID NO: 49 представляет собой праймер для амплификации SM2982.
SEQ ID NO: 50 представляет собой праймер для амплификации SM2982.
SEQ ID NO: 51 представляет собой зонд для SM2982.
SEQ ID NO: 52 представляет собой зонд для SM2982.
SEQ ID NO: 53 представляет собой праймер для амплификации SM2984.
SEQ ID NO: 54 представляет собой праймер для амплификации SM2984.
SEQ ID NO: 55 представляет собой зонд для SM2984.
SEQ ID NO: 56 представляет собой зонд для SM2984.
SEQ ID NO: 57 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды PZE01271951242 на хромосоме 1 маиса, 272937470 п. о. - 272938270 п. о. (интервал 8).
SEQ ID NO: 58 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды PZE0211924330 на хромосоме 2 маиса, 12023306 п. о. - 12024104 п. о. (интервал 9).
SEQ ID NO: 59 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды PZE03223368820 на хромосоме 3 маиса, 225037202 п. о. - 225038002 п. о. (интервал 10).
SEQ ID NO: 60 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды PZE03223703236 на хромосоме 3 маиса, 225340531 п. о. - 225341331 п. о. (интервал 11).
SEQ ID NO: 61 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды PZE05158466685 на хромосоме 5 маиса, 159120801 п. о. - 159121601 п. о. (интервал 12).
SEQ ID NO: 62 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды PZE0911973339 на хромосоме 9 маиса, 12104536 п. о. - 12105336 п. о. (интервал 13).
SEQ ID NO: 63 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды S_l8791654 на хромосоме 9 маиса в пределах п. о. 225343590 - 225340433 (интервал 14).
SEQ ID NO: 64 представляет собой нуклеотидную последовательность, которая ассоциирована с локусом оптимизации потребления воды S_20808011 на хромосоме 9 маиса в пределах п. о. 14764415 - 14765098 (интервал 15).
SEQ ID NO: 65 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом А локуса оптимизации потребления воды.
SEQ ID NO: 66 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом В локуса оптимизации потребления воды.
SEQ ID NO: 67 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом С локуса оптимизации потребления воды.
SEQ ID NO: 68 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом D локуса оптимизации потребления воды.
SEQ ID NO: 69 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом Е локуса оптимизации потребления воды.
SEQ ID NO: 70 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом F локуса оптимизации потребления воды.
SEQ ID NO: 71 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом G локуса оптимизации потребления воды.
SEQ ID NO: 72 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом Н локуса оптимизации потребления воды.
SEQ ID NO: 73 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом I локуса оптимизации потребления воды.
SEQ ID NO: 74 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом J локуса оптимизации потребления воды.
SEQ ID NO: 75 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом K локуса оптимизации потребления воды.
SEQ ГО NO: 76 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом L локуса оптимизации потребления воды.
SEQ ID NO: 77 представляет собой нуклеотидную последовательность, которая ассоциирована с гаплотипом М локуса оптимизации потребления воды.
ПОДРОБНОЕ ОПИСАНИЕ
Объекты, раскрытые в настоящем изобретении, предусматривают композиции и способы идентификации, отбора и/или получения растений маиса с повышенной засухоустойчивостью (также называемой в данном документе оптимизацией потребления воды), а также растения маиса, идентифицированные, отобранные и/или полученные с помощью способа по настоящему изобретению. Кроме того, объекты, раскрытые в настоящем изобретении, предусматривают растения и/или идиоплазмы маиса, имеющие в пределах своих геномов один или несколько маркеров, ассоциированных с повышенной засухоустойчивостью.
Для оценки значения хромосомных интервалов, локусов, генов или маркеров в условиях стресса, вызванного засухой, разнородную идиоплазму подвергали скринингу в условиях контролируемого полевого эксперимента, предусматривающих контрольную обработку с обильным поливом и обработку с ограниченным поливом. Цель обработки с обильным поливом состояла в том, чтобы убедиться, что вода не ограничивала продуктивность сельскохозяйственной культуры. Напротив, цель обработки с ограниченным поливом состояла в том, чтобы убедиться, что вода стала главным лимитирующим ограничением для урожайности зерна. Основные эффекты (например, обработка и генотип) и взаимодействия (например, генотип х обработка) можно было определять, когда две обработки применяли рядом друг с другом в полевых условиях. Помимо этого, можно было оценивать количественно фенотипы, связанные с засухой, для каждого генотипа в панели, обеспечивая тем самым возможность проводить ассоциации маркер-признак.
При осуществлении на практике способ обработки с ограниченным поливом может варьировать в широких пределах, в зависимости от идиоплазмы, подлежащей скринингу, типа почвы и климатических условий в месте произрастания, снабжения водой до сезона вегетации и снабжения водой во время сезона вегетации, при этом упомянуты только некоторые переменные. Сперва идентифицируют место произрастания, в котором количество осадков во время сезона вегетации является низким (для сведения к минимуму вероятности непредусмотренной подачи воды) и которое подходит для культивирования растений. Кроме того, может быть важно определить временные рамки стресса, вследствие чего параметр определяют так, чтобы убедиться, что имеет место согласованность скрининга в разные годы и в разных местоположениях. Также следует учитывать понимание интенсивности обработки или, в некоторых случаях, потери урожая, ожидаемые при обработке с ограниченным поливом. Выбор интенсивности обработки, которая является слишком слабой, может не позволить выявить генотипическую изменчивость. Выбор интенсивности обработки, которая является слишком сильной, может приводить к большой экспериментальной ошибке. После того как идентифицированы временные рамки стресса и описана интенсивность обработки, полив можно контролировать способом, который согласуется с этими параметрами. В случае данных, полученных в настоящей заявке, использовали надежные места произрастания для испытаний, которые контролировались в течение многих лет, включая такие переменные как тенденции погоды, типы почвы, уровни питательных веществ и т.д. Это обеспечивает возможность большей эффективности при выявлении фенотипов и впоследствии генотипов, обуславливающих повышенную урожайность и/или засухоустойчивость.
Не подразумевается, что настоящее описание является подробным перечнем всех различных способов, с помощью которых может быть реализовано настоящее изобретение, или всех признаков, которые можно добавить к настоящему изобретению. Например, признаки, проиллюстрированные в отношении одного варианта осуществления, могут быть включены в другие варианты осуществления, а признаки, проиллюстрированные в отношении конкретного варианта осуществления, могут быть удалены из этого варианта осуществления. Таким образом, в настоящем изобретении предполагается, что в некоторых вариантах осуществления настоящего изобретения можно исключить или опустить любой признак или комбинацию признаков, изложенных в данном документе. Кроме того, многочисленные вариации и дополнения к различным вариантам осуществления, предлагаемым в данном документе, будут очевидны для специалистов в данной области в свете настоящего раскрытия, которое не отступает от сути настоящего изобретения. Следовательно, следующие описания предназначены для иллюстрации некоторых конкретных вариантов осуществления настоящего изобретения, а не исчерпывающего определения всех их преобразований, комбинаций и вариаций.
Если не указано иное, все технические и научные термины, используемые в данном документе, имеют то же значение, которое обычно понятно обычному специалисту в данной области, к которой относится настоящее изобретение. Терминология, используемая в данном документе при описании настоящего изобретения, предназначена только для описания конкретных вариантов осуществления и не предназначена для ограничения настоящего изобретения.
Все публикации, заявки на патенты, патенты и другие ссылки, цитируемые в данном документе, включены с помощью ссылки во всей своей полноте для объяснения идей, относящихся к предложению и/или абзацу, в котором приведена данная ссылка. Ссылки на используемые в данном документе методики предназначены для обозначения методик, как правило, известных в данной области, включая изменения этих методик или замены на эквивалентные методики, которые будут очевидны специалистам в данной области.
Если контекст не указывает на иное, то специально предполагается, что различные признаки настоящего изобретения, описанные в данном документе, можно использовать в любой комбинации. Более того, в настоящем изобретении также предполагается, что в некоторых вариантах осуществления настоящего изобретения любой признак или комбинацию признаков, изложенных в данном документе, можно исключить или опустить. С целью иллюстрации, если в данном описании утверждается, что композиция содержит компоненты А, В и С, то это, в частности, предполагает, что любое из А, В или С или их комбинацию можно опустить и отклонить по отдельности или в любой комбинации.
I. Определения
Хотя следующие термины, как предполагается, хорошо понятны обычному специалисту в данной области, следующие определения приведены для облегчения пояснения объектов, раскрытых в настоящем изобретении.
Все используемые в данном документе технические и научные термины, если ниже не указано иное, предполагаются как имеющие такое же значение, которое обычно понятно обычному специалисту в данной области. Ссылки на используемые в данном документе методики предназначены для обозначения методик, как правило, известных в данной области, включая изменения этих методик или замены на эквивалентные методики, которые будут очевидны специалистам в данной области. Хотя следующие термины, как предполагается, хорошо понятны обычному специалисту в данной области, следующие определения приведены для облегчения пояснения объектов, раскрытых в настоящем изобретении.
Предполагается, что применяемые в описании настоящего изобретения и прилагаемой формуле изобретения формы единственного числа также включают формы множественного числа, если контекст явно не указывает на иное.
Используемый в данном документе "и/или" относится к любой и всем возможным комбинациям одного или нескольких соответствующих перечисленных элементов и охватывает их, а также к отсутствию комбинаций при интерпретации в качестве альтернативы ("или").
Если не указано иное, все числа, выражающие количества ингредиентов, условия реакции и т.д., используемые в настоящем описании и формуле изобретения, следует понимать как модифицированные во всех случаях выражением "приблизительно". Подразумевается, что выражение "приблизительно", используемое в данном документе при упоминании измеряемого значения, такого как величина массы, веса, времени, объема, концентрации или процентной доли, охватывает изменения от указанной величины, составляющие в некоторых вариантах осуществления ±20%, в некоторых вариантах осуществления ±10%, в некоторых вариантах осуществления ±5%, в некоторых вариантах осуществления ±1%, в некоторых вариантах осуществления ±0,5% и в некоторых вариантах осуществления ±0,1%, поскольку такие изменения подходят для осуществления раскрытых способов. Соответственно, если не указано обратное, числовые параметры, изложенные в настоящем описании и прилагаемой формуле изобретения являются приблизительными величинами, которые могут варьировать, в зависимости от требуемых свойств, которых стремятся достичь с помощью объектов, раскрытых в настоящем изобретении.
Используемые в данном документе фразы, такие как "от X и Y" и "от приблизительно X до Y", следует интерпретировать как включающие X и Y. Используемые в данном документе, фразы, такие как "от приблизительно X до Y" означают "от приблизительно X до приблизительно Y", и фразы, такие как "от приблизительно X до Y", означают "от приблизительно X до приблизительно Y."
Используемый в данном документе термин "содержат", "содержит" и "содержащий" указывает на присутствие указанных признаков, целых чисел, стадий, действий, элементов и/или компонентов, но не исключает присутствие или добавление одного или нескольких других признаков, целых чисел, стадий, действий, элементов, компонентов и/или их групп.
Используемая в данном документе переходная фраза "состоящий фактически из" означает, что объем пункта формулы изобретения следует интерпретировать как охватывающий определенные материалы или стадии, перечисленные в пункте формулы изобретения, а также такие, которые существенно не влияют на основную(-ые) и новую(-ые) характеристику(-и) заявляемого изобретения. Таким образом, предусмотрено, что термин "состоящий фактически из", когда он применяется в пункте формулы настоящего изобретения, не интерпретируется как эквивалентный термину "содержащий".
Используемый в данном документе термин "аллель" относится к одному из двух или более различных нуклеотидов или нуклеотидных последовательностей, которые встречаются в конкретном локусе хромосомы.
Используемый в данном документе термин "интервал между пылением и выметыванием пестичных столбиков" (ASI) относится к промежутку между тем, когда растение начинает сбрасывать пыльцу (пыление), и когда оно начинает производить пестичные столбики (женские органы размножения). Данные собирают из расчета на делянку. В некоторых вариантах осуществления этот интервал выражают в днях.
"Локус" представляет собой положение на хромосоме, где расположены ген, или маркер, или аллель. В некоторых вариантах осуществления локус может охватывать один или несколько нуклеотидов.
Используемые в данном документе термины "требуемый аллель", "целевой аллель", "причинный аллель" и/или "представляющий интерес аллель" применяются взаимозаменяемо для обозначения аллеля, ассоциированного с требуемым признаком (например, любого из аллелей, перечисленных в таблицах 1-7 или их тесно ассоциированных аллелей).
Используемая в данном документе фраза "ассоциированный с" относится к распознаваемой и/или анализируемой взаимосвязи между двумя объектами. Например, фраза "ассоциированный с признаком оптимизации потребления воды" относится к признаку, локусу, гену, аллелю, маркеру, фенотипу и т.д., или к их экспрессии, присутствие или отсутствие которых может влиять на степень, уровень и/или размер, при которых растет растение или его представляющая интерес часть, которые имеют признак оптимизации потребления воды. Соответственно, маркер является "ассоциированным с" признаком, если он сцеплен с ним и если наличие маркера является индикатором того, будут ли и/или в какой степени требуемый признак или форма признака будут проявляться у растения/в идиоплазме, содержащих маркер. Аналогичным образом маркер является "ассоциированным с" аллелем, если он сцеплен с ним и если наличие маркера является индикатором того, присутствует ли аллель в растении/идиоплазме, содержащих маркер. Например, "маркер, ассоциированный с повышенной засухоустойчивостью" относится к маркеру, присутствие или отсутствие которого можно применять для предсказания того, будет ли и/или в какой степени растение будет демонстрировать фенотип засухоустойчивости (например, все маркеры, идентифицированные в таблицах 1-7, являются тесно ассоциированными с повышенной урожайностью маиса как в условиях засухи, так и в условиях, отличных от засухи).
Используемые в данном документе термины "возвратное скрещивание" и "проведение возвратного скрещивания" относятся к способу, при котором растение-потомок подвергают возвратному скрещиванию с одним из его родителей один или несколько раз (например, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или больше). В схеме возвратного скрещивания "родитель-донор" относится к родительскому растению с требуемым геном или локусом, подлежащими интрогрессии. "Родитель-реципиент" (используемый один или несколько раз) или "рекуррентный родитель" (используемый два или более раз) относится к родительскому растению в которое, ген или локус интрогрессируется. Например, см. Ragot, М. et al. Marker-assisted Backcrossing: A Practical Example, в TECHNIQUES ET UTILISATIONS DES MARQUEURS MOLECULAIRES LES COLLOQUES, Vol. 72, pp. 45-56 (1995); и Openshaw et al., Marker-assisted Selection in Backcross Breeding, in PROCEEDINGS OF THE SYMPOSIUM "ANALYSIS OF MOLECULAR Marker Data," pp. 41-43 (1994). Первое скрещивание дает начало поколению F1. Термин "ВС1" относится к второму использованию рекуррентного родителя, "ВС2" относится к третьему использованию рекуррентного родителя и т.д. В некоторых вариантах осуществления количество возвратных скрещиваний может составлять от приблизительно 1 до приблизительно 10 (например, 1, 2, 3, 4, 5, 6, 7, 8, 9 и 10). В некоторых вариантах осуществления количество возвратных скрещиваний составляет приблизительно 7.
Используемые в данном документе термины "скрещивание" или "подвергнутый скрещиванию" относятся к слиянию гамет в результате опыления с получением потомства (например, клеток, семян или растений). Данный термин охватывает как половое скрещивание (опыление одного растения другим), так и самоопыление (гомоклинное опыление, например, если пыльца и семязачаток происходят от одного и того же растения). Термин "скрещивание" относится к событию слияния гамет в результате опыления с образованием потомства.
Используемые в данном документе термины "культивар" и "сорт" относятся к группе похожих растений, которые можно отличать от других сортов в пределах одного вида по структурным или генетическим признакам и/или характеристикам.
Используемые в данном документе термины "элитный" и/или "элитная линия" относятся к любой линии, которая является практически гомозиготной и получена в результате селекции и отбора по требуемым агрономическим характеристикам.
Используемые в данном документе термины "экзотический", "экзотическая линия" и "экзотическая идиоплазма" относятся к любому растению, линии или идиоплазме, которые не являются элитными. В общем случае экзотические растения/идиоплазмы не происходят от любого известного элитного растения или идиоплазмы, а отобраны для введения одного или нескольких требуемых генетических элементов в селекционную программу (например, для введения новых аллелей в селекционную программу).
"Контроль", или "контрольное растение", или "контрольная растительная клетка" обеспечивают эталонную точку для измерения изменений в фенотипе заявленного растения или растительной клетки. Контрольное растение или растительная клетка могут включать, например: (а) растение или клетку дикого типа, т.е. имеющих такой же генотип, что и у исходного материала для генетического изменения (например, интрогрессии), которое привело к заявленному растению или клетке; (b) растение или растительную клетку, имеющих такой же генотип, что и у исходного материала, но которые были подвергнуты трансформации нулевой конструкцией (т.е. конструкцией, которая не экспрессирует специфический белок транспортной клетки и переносчик сахаров, описываемые в данном документе); (с) растение или растительную клетку, которые являются нетрансформированными сегрегантами среди потомства заявленного растения или растительной клетка или; (d) растение, которое фактически идентично в большинстве аспектов заявленному растению или растительной клетке, однако отличается генотипом, в частности SNP, гаплотипом, имеет вставку/делецию (например, контрольное растение маиса, имеющее неблагоприятный аллель в специфическом положении хромосомы, в сопоставлении с заявленным (экспериментальным) растением маиса с благоприятным аллелем в том же положении).
Используемый в данном документе термин "хромосома" применяется в своем принятом в данной области значении, обозначающем самореплицирующуюся генетическую структуру в клеточном ядре, содержащую клеточную ДНК и несущую в своей нуклеотидной последовательности линейный массив из генов. Номера хромосом Zea mays, раскрытые в данном документе, относятся к таким, которые изложены в Perin et al., 2002, которые связаны с эталонной системой номенклатуры, адаптированной Национальным институтом сельскохозяйственных исследований (INRA; Париж, Франция).
Используемая в данном документе фраза "консенсусная последовательность" относится к последовательности ДНК, построенной для идентификации различий нуклеотидов (например, SNP и полиморфизмов вставок/делеций) у аллелей в локусе. Консенсусная последовательность может представлять собой одну из нитей ДНК в локусе и определять нуклеотид(ы) в одном или нескольких положениях (например, в одном или нескольких SNP и/или в одной или нескольких вставках/делециях) в локусе. В некоторых вариантах осуществления консенсусную последовательность используют для разработки олигонуклеотидов и зондов для выявления полиморфизмов в локусе.
"Генетическая карта" представляет собой описание взаимосвязей при генетическом сцеплении между локусами на одной или нескольких хромосомах в пределах данного вида, обычно изображаемое в форме схемы или таблицы. Для каждой генетической карты расстояния между локусами измеряют по значениям частоты рекомбинации между ними. Рекомбинацию между локусами можно выявлять с помощью ряда маркеров. Генетическая карта является продуктом, получаемым с учетом картирующей популяции, типов применяемых маркеров и потенциала полиморфизма каждого маркера среди различных популяций. Порядок расположения локусов и генетические расстояния между ними могут отличаться на разных генетических картах.
Используемый в данном документе, термин "генотип" относится к генетической конституции особи (или группы особей) в одном или нескольких генетических локусах, в противоположность наблюдаемому, и/или выявляемому, и/или проявляемому признаку (фенотипу). Генотип определяют по аллелю(-ям) одного или нескольких известных локусов, которые особь наследует от своих родителей. Термин "генотип" можно использовать для обозначения генетической конституции особи по одному локусу, по нескольким локусам, или, в более широком смысле, термин "генотип" можно использовать для обозначения генетического строения особи по всем генам в его геноме. Генотипы можно характеризовать опосредованно, например, с применением маркеров, и/или характеризовать непосредственно путем, например, секвенирования нуклеиновых кислот.
Используемый в данном документе термин "идиоплазма" относится к генетическому материалу особи (например, растения), группы особей (например, линии, сорта или семейства растений) или клона, происходящего из линии, сорта, вида или культуры, или полученному из них. Идиоплазма может представлять собой часть организма или клетку, или может быть отделена от организма или клетки. Как правило, в идиоплазме представлен генетический материал со специфическим генетическим строением, который обеспечивает основу для некоторых или всех наследственных качеств организма или культуры клеток. Используемый в данном документе термин "идиоплазма" включает клетки, семена или ткани, из которых можно выращивать новые растения, а также части растений, из которых можно культивировать целое растение (например, листья, стебли, почки, корни, пыльцу, клетки и т.д.). В некоторых вариантах осуществления идиоплазма включает без ограничения культуру тканей.
"Гаплотип" представляет собой генотип особи по множеству генетических локусов, т.е. комбинацию аллелей. Как правило, генетические локусы, которые определяют гаплотип, являются физически и генетически сцепленными, т.е. расположены в одном сегменте хромосомы. Термин "гаплотип" может относится к полиморфизмам в конкретном локусе, таком как одиночный маркерный локус, или полиморфизмам в множественных локусах вдоль сегмента хромосомы (например, гаплотип может составлять любая комбинация по меньшей мере из двух аллелей, перечисленных в таблице 1, 2, 3, 4, 5, 6 или 7 соответственно).
Используемый в данном документе термин "гетерозиготный" относится к генетическому статусу, при котором в соответствующих локусах на гомологичных хромосомах расположены разные аллели. В некоторых вариантах осуществления родительская линия или растение-потомок маиса являются гетерозиготными по любому из аллелей урожайности 1-7
Используемый в данном документе термин "гомозиготный" относится к генетическому статусу, при котором в соответствующих локусах на гомологичных хромосомах расположены идентичные аллели. В некоторых вариантах осуществления родительская линия или растение-потомок маиса являются гомозиготными по любому из аллелей урожайности 1-7
Используемый в данном документе в контексте селекции растений термин "гибридный" относится к растению, которое является потомком генетически разнородных родителей, полученным за счет скрещивания растений различных линий, или селекционных сортов, или видов, включая без ограничения скрещивание между двумя инбредными линиями.
Используемый в данном документе термин "инбредный" относится к практически гомозиготному растению или сорту Термин может относится к растению или сорту растения, которые являются практически гомозиготными на протяжении всего генома, или которые являются практически гомозиготными в части генома, которая представляет особый интерес.
Используемые в данном документе термины "интрогрессия", "осуществление интрогрессии" и "подвергнутый интрогрессии" относятся как к естественному, так и к искусственному переносу требуемого аллеля или комбинации требуемых аллелей генетического локуса или генетических локусов из одного генетического окружения в другое. Например, требуемый аллель в определенном локусе может быть перенесен по меньшей мере в одного потомка посредством полового скрещивания двух родителей одного и того же вида, при котором по меньшей мере один из родителей имеет в своем геноме требуемый аллель. Например, в качестве альтернативы перенос аллеля может происходить посредством рекомбинации между двумя донорными геномами, например, в слитом протопласте, где по меньшей мере один из донорных протопластов имеет в своем геноме требуемый аллель. Требуемый аллель может представлять собой отобранный аллель маркера, QTL, трансгена или им подобные. Потомство, содержащее требуемый аллель, может быть подвергнуто возвратному скрещиванию один или несколько раз (например, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более раз) с линией, имеющей требуемое генетическое окружение, с отбором по требуемому аллелю, в результате чего требуемый аллель встраивается в требуемое генетическое окружение. Например, маркер, ассоцированный с засухоустойчивостью (например, любые маркеры, показанные в таблицах 1-7), можно интрогрессировать из донора в рекуррентного родителя, который является чувствительным к засухе. Полученное потомство затем можно подвергать возвратному скрещиванию один или несколько раз и подвергать отбору до тех пор, пока потомок не будет содержать генетический маркер(-ы), ассоциированные с засухоустойчивостью в генетическом окружении рекуррентного родителя.
Используемый в данном документе термин "сцепление" относится к явлению, при котором аллели на одной и той же хромосоме в большинстве случае передаются вместе более часто, чем ожидается в том случае, если бы их передача была независимой. Таким образом, говорится, что два аллеля на одной и той же хромосоме являются "сцепленными", когда в следующем поколении они сегрегируют друг от друга в некоторых вариантах осуществления менее чем в 50% случаев, в некоторых вариантах осуществления менее чем в 25% случаев, в некоторых вариантах осуществления менее чем в 20% случаев, в некоторых вариантах осуществления менее чем в 15% случаев, в некоторых вариантах осуществления менее чем в 10% случаев, в некоторых вариантах осуществления менее чем в 9% случаев, в некоторых вариантах осуществления менее чем в 8% случаев, в некоторых вариантах осуществления менее чем в 7% случаев, в некоторых вариантах осуществления менее чем в 6% случаев, в некоторых вариантах осуществления менее чем в 5% случаев, в некоторых вариантах осуществления менее чем в 4% случаев, в некоторых вариантах осуществления менее чем в 3% случаев, в некоторых вариантах осуществления менее чем в 2% случаев и в некоторых вариантах осуществления менее чем в 1% случаев.
Соответственно, "сцепление" обычно предполагает и также может относится к физическому соседству на хромосоме. Таким образом, два локуса являются сцепленными, если они находятся в пределах в некоторых вариантах осуществления 20 сантиморганид (сМ), в некоторых вариантах осуществления 15 сМ, в некоторых вариантах осуществления 12 сМ, в некоторых вариантах осуществления 10 сМ, в некоторых вариантах осуществления 9 сМ, в некоторых вариантах осуществления 8 сМ, в некоторых вариантах осуществления 7 сМ, в некоторых вариантах осуществления 6 сМ, в некоторых вариантах осуществления 5 сМ, в некоторых вариантах осуществления 4 сМ, в некоторых вариантах осуществления 3 сМ, в некоторых вариантах осуществления 2 сМ и в некоторых вариантах осуществления 1 сМ друг от друга. Аналогичным образом локус урожайности (например, аллели урожайности 1-8) у объектов, раскрытых в настоящем изобретении, сцеплен с маркером (например, генетическим маркером), если в некоторых вариантах осуществления он находится в пределах 20, 15, 12, 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1 сМ от маркера. Таким образом, маркер, сцепленный с любым из аллелей урожайности 1-8, можно использовать для отбора, идентификации или получения растений маиса с повышенной устойчивостью к засухе и/или повышенной урожайностью.
В некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, предпочтительно определить установленный диапазон сцепления, например, от приблизительно 10 сМ до приблизительно 20 сМ, от приблизительно 10 сМ до приблизительно 30 сМ, или от приблизительно 10 сМ до приблизительно 40 сМ. Чем более тесно маркер сцеплен со вторым локусом (например, аллелями урожайности 1-8), тем лучшим индикатором для второго локуса становится такой маркер. Таким образом, "тесно сцепленные" или, как используется взаимозаменяемо, "тесно ассоциированные" локусы или маркеры, такие как маркерный локус и второй локус, проявляют частоту межлокусной рекомбинации, составляющую приблизительно 10%, 9%, 8%, 7%, 6%, 5%, 4%, 3% или 2% или меньше. В некоторых вариантах осуществления соответствующие локусы проявляют частоту рекомбинации, составляющую приблизительно 1% или меньше, например, приблизительно 0,75%, 0,5%, 0,25% или меньше. Также может считаться, что два локуса, которые локализованы на одной и той же хромосоме, и, следовательно, расстояние, при котором рекомбинация между двумя локусами происходит при частоте, составляющей менее приблизительно 10% (например, приблизительно 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2%, 1%, 0,75%, 0,5% или 0,25% или меньше), находятся "по соседству" друг с другом. Поскольку одна сМ представляет собой расстояние между двумя маркерами, которые демонстрируют частоту рекомбинации в 1%, любой маркер является тесно сцепленным (генетически и физически) с любым другим маркером, который находится по соседству с ним, например, на расстоянии приблизительно 10 сМ или меньше. Два тесно сцепленных маркера на одной и той же хромосоме могут быть расположены на расстоянии приблизительно 9, 8, 7, 6, 5, 4, 3, 2, 1, 0,75, 0,5 или 0,25 сМ или меньше друг от друга. Сантиморганида ("сМ") или единица генетической карты (m. u.) представляет собой единицу измерения частоты рекомбинации и определяется как расстояние между генами, при котором один продукт мейоза из 100 является рекомбинантным. Одна сМ соответствует 1% вероятности того, что маркер в одном генетическом локусе будет отделятся от маркера во втором локусе в результате кроссинговера за одно поколение. Таким образом, частота рекомбинации (RF), соответствующая 1%, эквивалентна 1 m. u.
Используемая в данном документе фраза "группа сцепления" относится ко всем генам или генетическим признакам, которые расположены на одной и той же хромосоме. В пределах группы сцепления локусы, расположенные достаточно близко друг к другу, могут проявлять сцепление при генетических скрещиваниях. Поскольку вероятность кроссинговера возрастает по мере увеличения физического расстояния между локусами в хромосоме, то локусы, местоположения которых находятся далеко друг от друга в пределах группы сцепления, могут не проявлять какого-либо выявляемого сцепления в прямых генетических тестах. Термин "группа сцепления" используется преимущественно для обозначения генетических локусов, которые проявляют сцепленное поведение в генетических системах, в которых соотнесения с хромосомами еще не были выполнены. Таким образом, термин "группа сцепления" является синонимом физического объекта хромосомы, хотя обычному специалисту в данной области будет понятно, что группа сцепления также может быть определена как соответствующая участку (т.е. части, меньше чем целая) данной хромосомы или, например, любому из интервалов 1-15, определяемых в данном документе
Используемый в данном документе термин "неравновесное сцепление" или "LD" относится к неслучайной сегрегации генетических локусов или признаков (или обоих). В любом случае неравновесное сцепление означает, что соответствующие локусы находятся в достаточном физическом соседстве вдоль длины хромосомы, так что они сегрерируют вместе с частотой, превышающей частоту при случайной сегрегации (т.е. с частотой, соответствующей неслучайной сегрегации) (в случае косегрегирующих признаков локусы, которые лежат в основе данных признаков, находятся в достаточном соседстве друг от друга). Маркеры, которые демонстрируют неравновесное сцепление, считаются сцепленными. Сцепленные локусы косегрегируют в более чем 50% случаев, например, от приблизительно 51% до приблизительно 100% случаев. Другими словами, два косегрегирующих маркера характеризуются частотой рекомбинации, составляющей менее 50% (и по определению разделены менее чем 50 сМ на одной и той же хромосоме). Используемое в данном документе сцепление может иметь место между двумя маркерами или, в качестве альтернативы, между маркером и фенотипом. Маркерный локус может быть "ассоциирован с" (сцеплен с) признаком, например, засухоустойчивостью. Степень сцепления генетического маркера с фенотипическим признаком измеряют, например, в виде статистической вероятности косегрегации этого маркера с фенотипом.
Неравновесное сцепление чаще всего оценивают с применением показателя r2, который рассчитывают с помощью формулы, описанной в Hill and Robertson, Theor. Appl. Genet. 38:226 (1968). Если r2 = 1, между двумя маркерными локусами наблюдается полное неравновесное сцепление, а это означает, что данные маркеры не разделяются при рекомбинации и характеризуются одинаковой частотой аллелей. Значения r2, превышающие 1/3, указывают на достаточно сильное неравновесное сцепление, которое применимо для картирования. Ardlie et al., Nature Reviews Genetics 3:299 (2002). Поэтому, аллели находятся в неравновесном сцеплении, если значения r2 между парными маркерными локусами превышают или равны приблизительно 0,33, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или 1,0.
Используемый в данном документе термин "равновесное сцепление" описывает ситуацию, при которой два маркера сегрегируют независимо, т.е. распределяются среди потомства случайным образом. Маркеры, которые демонстрируют равновесное сцепление, считаются несцепленными (независимо от того, лежат ли они на одной и той же хромосоме или нет).
Используемые в данном документе термины "маркер", "генетический маркер", "нуклеиновая кислота-маркер" и "молекулярный маркер" используются взаимозаменяемо для обозначения идентифицируемого положения на хромосоме, наследование которого можно отслеживать, и/или реагента, который применяют в способах для визуализации отличий в последовательностях нуклеиновой кислоты, присутствующих в таких идентифицируемых положениях на хромосомах. Таким образом, в некоторых вариантах осуществления маркер содержит известную или выявляемую последовательность нуклеиновой кислоты. Примеры маркеров включают без ограничения генетические маркеры, состав белков, уровни пептидов, уровни белков, состав масел, уровни масел, состав углеводов, уровни углеводов, состав жирных кислот, уровни жирных кислот, состав аминокислот, уровни аминокислот, биполимеры, состав крахмалов, уровни крахмалов, ферментируемый крахмал, выход ферментации, эффективность ферментации (например, регистрируемый как перевариваемость через 24, 48 и/или 72 часа), выход энергии, вторичные соединения, метаболиты, морфологические характеристики и агрономические характеристики. Соответственно, маркер может предусматривать нуклеотидную последовательность, которая была ассоциирована с представляющим интерес аллелем или аллелями и которая указывает на присутствие или отсутствие представляющего интерес аллеля или аллелей в клетке или организме, и/или реагент, который применяют для визуализации отличий в нуклеотидной последовательности в таком идентифицируемом положении или положениях. Маркер может представлять собой без ограничения аллель, ген, гаплотип, полиморфизм длин рестрикционных фрагментов (RFLP), простой повтор последовательности (SSR), случайно амплифицированную полиморфную ДНК (RAPD), рестрикционный полиморфизм амплифицированных последовательностей (CAPS) (Rafalski and Tingey, Trends in Genetics 9:275 (1993)), полиморфизм длин амплифицированных фрагментов (AFLP) (Vos et al., Nucleic Acids Res. 23:4407 (1995)), однонуклеотидный полиморфизм (SNP) (Brookes, Gene 234:177 (1993)), амплифицированную область, охарактеризованную последовательностью (SCAR) (Paran and Michelmore, Theor. Appl. Genet. 85:985 (1993)), ДНК-маркирующий сайт (STS) (Onozaki et al., Euphytica 138:255 (2004)), однонитевой конформационный полиморфизм (SSCP) (Orita et al., Proc. Natl. Acad. Sci. USA 86:2766 (1989)), последовательность, расположенную внутри простых повторов последовательности (ISSR) (Blair et al., Theor. Appl. Genet. 98:780 (1999)), полимофизм последовательности, амплифицируемой между ретротранспозонами (IRAP), полимофизм последовательности, амплифицируемой между ретротранспозоном и микросателлитом (REMAP) (Kalendar et al., Theor. Appl. Genet. 98:704 (1999)) или продукт расщепления РНК (такой как метка Lynx). Маркер может присутствовать в геномной или экспрессируемой нуклеиновых кислотах (например, EST). Термин маркер также может относится к нуклеиновым кислотам, используемым в качестве зондов или праймеров (например, пар праймеров) для применения в амплификации, гибридизации и/или выявлении молекул нуклеиновой кислоты в соответствии со способами, хорошо известными в данной области. Огромное число молекулярных маркеров маиса известны в данной области и опубликованы или доступны из различных источников, таких как интернет-ресурс Maize GDB и интернет-ресурс Arizona Genomics Institute, обслуживаемый Аризонским университетом.
В некоторых вариантах осуществления маркер соответствует продукту амплификации, полученному путем амплификации нуклеиновой кислоты Zea mays с помощью одного или нескольких олигонуклеотидов, например, с помощью полимеразной цепной реакции (ПЦР). Используемая в данном документе в контексте маркера фраза "соответствует продукту амплификации" относится к маркеру, который имеет нуклеотидную последовательность, которая является такой же (допускаются мутации, введенные за счет реакции амплификации самой по себе, и/или встречающиеся в природе и/или искусственные аллельные отличия), как и у продукта амплификации, который получен за счет амплификации геномной ДНК Zea mays с помощью конкретного набора олигонуклеотидов. В некоторых вариантах осуществления амплификация осуществляется посредством ПЦР, а олигонуклеотиды представляют собой праймеры для ПЦР, которые разработаны для гибридизации с противоположными нитями геномной ДНК Zea mays, чтобы амплифицировать последовательность геномной ДНК Zea mays, находящуюся между последовательностями, с которыми гибридизируются праймеры для ПЦР в геномной ДНК Zea mays. Амплифицированный фрагмент, который получен в результате одного или нескольких раундов амплификации с применением такой конфигурации праймеров, представляет собой двухнитевую нуклеиновую кислоту, одна нить которой имеет нуклеотидную последовательность, которая в направлении от 5' к 3' содержит последовательность одного из праймеров, последовательность геномной ДНК Zea mays, расположенной между праймерами, и обратно комплементарную последовательность второго праймера. Как правило, "прямой" праймер задается как праймер, который имеет такую же последовательность, что и подпоследовательность (произвольно заданная) "главной" нити двухнитевой нуклеиновой кислоты, подлежащей амплификации, вследствие чего "главная" нить амплифицированного фрагмента включает в направлении от 5' к 3' нуклеотидную последовательность, которая равна последовательности прямого праймера - последовательность, расположенную между прямым и обратным праймерами главной нити геномного фрагмента - обратно комплементарную последовательность обратного праймера. Соответственно, маркер, который "соответствует" амплифицированному фрагменту, представляет собой маркер, который имеет такую же последовательность, как и одна из нитей амплифицированного фрагмента.
Маркеры, соответствующие генетическим полиморфизмам между представителями популяции, можно выявлять с помощью способов, общепризнанных в данной области. Они включают, например, секвенирование нуклеиновой кислоты, способы гибридизации, способы амплификации (например, способы специфической амплификации последовательности на основе ПЦР), выявление полиморфизмов длин рестрикционных фрагментов (RFLP), выявление изоферментых маркеров, выявление полинуклеотидных полиморфизмов при помощи аллель-специфической гибридизации (ASH), выявление амплифицированных вариабельных последовательностей генома растения, выявление самоподдерживающейся системы репликации последовательностей, выявление простых повторов последовательности (SSR), выявление однонуклеотидных полиморфизмов (SNP) и/или выявление полиморфизмов длин амплифицированных фрагментов (AFLP). Также известны общепринятые способы выявления экспрессируемых последовательностей-меток (EST) и маркеров SSR, происходящих от последовательностей EST и случайно амплифицированной полиморфной ДНК (RAPD).
Используемая в данном документе фраза "анализ на основе маркеров" относится к способу выявления полиморфизма в конкретном локусе с применением конкретного способа, такого как без ограничения измерение по меньшей мере одного фенотипа (такого как цвет семени, содержание масел или визуально выявляемый признак); анализы на основе нуклеиновой кислоты, включая без ограничений анализ полиморфизма длин рестрикционных фрагментов (RFLP), удлинение нити на одно основание, электрофорез, выравнивание последовательностей, аллель-специфическая гибридизация олигонуклеотидов (ASO), анализ случайно амплифицированной полиморфной ДНК (RAPD), технологии на основе микрочипов, анализы TAQMAN®, анализы с тестами ILLUMINA® GOLDENGATE®, технологии секвенирования нуклеиновой кислоты; анализы пептидов и/или полипептидов; или любая другая методика, которую можно использовать для выявления полиморфизма у организма в представляющем интерес локусе. Соответственно, в некоторых вариантах осуществления настоящего изобретения маркер выявляют с помощью амплификации нуклеиновой кислоты Zea mays с помощью двух олигонуклеотидных праймеров, например, посредством реакции амплификации, такой как полимеразная цепная реакция (ПЦР).
"Маркерный аллель", "аллель", также описываемый как "аллель маркерного локуса", может относится к одной из множества полиморфных нуклеотидных последовательностей, находящихся в маркерном локусе в популяции, которая является полиморфной по данному маркерному локусу.
"Отбор с помощью маркеров" (MAS) представляет собой способ, при котором фенотипы отбирают на основании маркерных генотипов. Отбор с помощью маркеров включает применение маркерных генотипов для идентификации растений, подлежащих включению в селекционную программу и/или исключению из нее или высаживанию.
"Противоотбор с помощью маркеров" представляет собой способ, при котором маркерные генотипы применяют для идентификации растений, которые не будут отбираться, обеспечивая возможность их удаления из селекционной программы или высаживания. Таким образом, в селекционных программах для растения маиса может применяться любая информация, перечисленная в таблицах 1-7, чтобы проводить противообор с помощью маркеров для исключения линий или идиоплазмы маиса, которые не характеризуются повышенной засухоустойчивостью.
Используемые в данном документе термины "маркерный локус", "локус", "локусы" и "маркерные локусы" относятся к специфическому местоположению или местоположению на хромосоме в геноме организма, в которых может находиться специфический маркер или маркеры. Маркерный локус можно применять для отслеживания присутствия второго сцепленного локуса, например, сцепленного локуса, который кодирует фенотипический признак или вносит свой вклад в его экспрессию. Например, маркерный локус можно применять для мониторинга сегрегации аллелей в локусе, в таком как QTL или отдельный ген, которые генетически или физически сцеплены с маркерным локусом.
Используемый в данном документе термин "зонд" или "молекулярный зонд" относится к однонитевой олигонуклеотидной последовательности, которая будет образовывать стабилизированный водородными связями дуплекс с комплементарной последовательностью в целевой аналите на основе последовательности нуклеиновой кислоты или ее кДНК-производной. Таким образом, термины "маркерный зонд" и "зонд" относятся к нуклеотидной последовательности или молекуле нуклеиновой кислоты, которые можно применять для выявления присутствия одного или нескольких конкретных аллелей в пределах маркерного локуса (например, к зонду на основе нуклеиновой кислоты, который комплементарен всему или части маркера или маркерного локуса) посредством гибридизации нуклеиновых кислот. Маркерные зонды, содержащие приблизительно 8, 10, 15, 20, 30, 40, 50, 60, 70, 80, 90, 100 или более смежных нуклеотидов, можно применять для гибридизации нуклеиновых кислот. В качестве альтернативы в некоторых аспектах маркерный зонд относится к зонду любого типа, способному различать (т.е. генотипировать) конкретный аллель, присутствующий в маркерном локусе. Неограничивающие примеры зонда по настоящему изобретению включают SEQ ID NO: 27, SEQ ID NO: 28, SEQ ID NO: 31, SEQ ID NO: 32, SEQ ID NO: 35, SEQ ID NO: 36, SEQ ID NO: 39, SEQ ID NO: 40, SEQ ID NO: 43, SEQ ID NO: 44, SEQ ID NO: 47, SEQ ID NO: 48, SEQ ID NO: 51, SEQ ID NO: 52, SEQ ID NO: 55 и/или SEQ ID NO: 56, а также последовательности, находящиеся в таблицах 1-7.
Используемый в данном документе термин "молекулярный маркер" можно использовать для обозначения генетического маркера, как определено выше, или кодируемого им продукта (например, белка), применяемого в качестве эталона при идентификации сцепленного локуса. Молекулярный маркер может происходить из геномных нуклеотидных последовательностей или из экспрессируемых нуклеотидных последовательностей (например, из сплайсированной РНК, кДНК и т.д.). Данный термин также относится к нуклеотидным последовательностям, комплементарным маркерным последовательностям или фланкирующим их, таким как нуклеотидные последовательности, применяемые в качестве зондов и/или праймеров, способных амплифицировать маркерную последовательность. Нуклеотидные последовательности являются "комплементарными", если они специфически гибридизируются в растворе, например, согласно правилам спаривания оснований Уотсона-Крика. Некоторые маркеры, описанные в данном документе, также могут обозначаться как маркеры гибридизации, если они расположены в области вставки/делеции. Это обусловлено тем, что область вставки по определению является полиморфизмом по отношению к растению без вставки. Таким образом, маркер нужен лишь для указания того, присутствует ли область вставки/делеции или отсутствует. Для идентификации такого маркера гибридизации можно применять любую подходящую технологию выявления маркера, например, технологию для выявления SNP.
Используемый в данном документе термин "праймер" относится к олигонуклеотиду, который способен отжигаться с нуклеиновой кислотой-мишенью и служить в качестве точки начала синтеза ДНК, будучи помещенным в условия, в которых индуцируется синтез продукта удлинения праймера (например, в присутствии нуклеотидов и средства для осуществления полимеризации, такого как ДНК-полимераза, и при подходящих температуре и показателе рН). Праймер (в некоторых вариантах осуществления праймер для удлинения и в некоторых вариантах осуществления праймер для амплификации) в некоторых вариантах осуществления является однонитевым для максимальной эффективности удлинения и/или амплификации. В некоторых вариантах осуществления праймер представляет собой олигодезоксирибонуклеотид. Праймер, как правило, является достаточно длинным, чтобы служить затравкой для синтеза продуктов удлинения и/или амплификации в присутствии средства для полимеризации. Минимальная длина праймера может зависеть от многих факторов, в том числе без ограничения от температуры и состава (содержания А/Т по сравнению с G/C) праймера. В контексте праймеров для амплификации, они, как правило, представлены в виде пары из двунаправленных праймеров, состоящей из одного прямого и одного обратного праймера, или представлены в виде пары из прямых праймеров, обычно применяемых в области амплификации ДНК, как, например, при ПЦР-амплификации. Соответственно, будет понятно, что используемый в данном документе термин "праймер" может относиться более чем к одному праймеру, в частности, в случае, когда существует некоторая неясность в информации о концевой последовательности(-ях) целевой области, подлежащей амплификации. Следовательно, "праймер" может включать совокупность праймерных олигонуклеотидов, содержащих последовательности, представляющие возможные вариации в последовательности, или он содержит нуклеотиды, обеспечивающие возможность обычного спаривания оснований.
Праймеры можно получить любым подходящим способом. Способы получения олигонуклеотидов с конкретной последовательностью известны из в данной области и включают, например, клонирование и рестрикцию соответствующих последовательностей, а также прямой химический синтез. Способы химического синтеза могут включать, например, фосфодиэфирный или фосфотриэфирный способ, диэтилфосфорамидатный способ и способ с использованием твердых подложек, раскрытый в патенте США №4458066. При необходимости праймеры можно пометить путем включения в их состав выявляемых фрагментов, например, спектроскопических, флуоресцентных, фотохимических, биохимических, иммунохимических или химических фрагментов.
Неограничивающие примеры праймеров по настоящему изобретению включают SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 29, SEQ ID NO: 30, SEQ ID NO: 33, SEQ ID NO: 34, SEQ ID NO: 37, SEQ ID NO: 38, SEQ ID NO: 41, SEQ ID NO: 42, SEQ ID NO: 45, SEQ ID NO: 46, SEQ ID NO: 49, SEQ ID NO: 50, SEQ ID NO: 53 и/или SEQ ID NO: 54. Способ ПЦР хорошо описан в руководствах и известен специалисту в данной области. После амплификации путем ПЦР целевые полинуклеотиды можно выявлять посредством гибридизации с полинуклеотидным зондом, который образует стабильный гибрид с целевой последовательностью в условиях гибридизации и отмывки от жестких до умеренно жестких. Если ожидается, что зонды будут фактически полностью комплементарны (т.е. на приблизительно 99% или больше) целевой последовательности, можно применять жесткие условия. Если ожидается некоторое несовпадение, например, если ожидается наличие вариантов штаммов, вследствие чего зонд не будет полностью комплементарен, то жесткость гибридизации можно уменьшить. В некоторых вариантах осуществления условия выбирают для исключения неспецифического/случайного связывания. Условия, которые влияют на гибридизацию, и которые выбирают для предупреждения неспецифического связывания, известны из уровня техники и описаны, например, в Sambrook & Russell (2001). Molecular Cloning: A Laboratory Manual. Third Edition. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York, United States of America. В целом, более низкая концентрация солей и более высокая температура гибридизации и/или отмывок повышают жесткость условий гибридизации.
Различные нуклеотидные последовательности или полипептидные последовательности, характеризующиеся гомологией, обозначаются в данном документе как "гомологи" или "гомологичные". Термин гомолог включает гомологичные последовательности от одного и того же и другого вида и ортологичные последовательности от одного и того же и другого вида. "Гомология" относится к уровню сходства двух или более нуклеотидных последовательностей и/или аминокислотных последовательностей, выраженному как процент идентичности положений (т.е. сходства или идентичности последовательностей). Гомология также относится к концепции сходных функциональных свойств различных нуклеиновых кислот, аминокислот и/или белков.
Используемая в данном документе фраза "гомология нуклеотидных последовательностей" относится к наличию гомологии между двумя последовательностями. Полинуклеотиды имеют "гомологичные" последовательности, если последовательность нуклеотидов в двух последовательностях является одинаковой при выравнивании для максимального соответствия. "Процентное значение гомологии последовательностей" для полинуклеотидов, такое как 50-, 55-, 60-, 65-, 70-, 75-, 80-, 85-, 90-, 95-, 96-, 97-, 98-, 99- или 100-процентная гомология последовательностей, можно определять путем сравнения двух оптимально выровненных последовательностей на протяжении окна сравнения (например, размером приблизительно 20-200 смежных нуклеотидов), где часть полинуклеотидной последовательности в окне сравнения может включать добавления или делеции (т.е. гэпы) по сравнению с эталонной последовательностью для оптимального выравнивания двух последовательностей. Оптимальное выравнивание последовательностей для сравнения можно осуществлять с помощью компьютерных реализаций известных алгоритмов или путем визуального просмотра. Общедоступными алгоритмами сравнения последовательностей и множественного выравнивания последовательностей являются соответственно программы средства поиска основного локального выравнивания (BLAST; Altschul et al. (1990) J Mol Biol 215:403-10; Altschul et al. (1997) Nucleic Acids Res 25:3389-3402) и ClustalX (Chenna et al. (2003) Nucleic Acids Res 31:3497-3500), обе из которых доступны в Интернете. Другие подходящие программы включают без ограничения GAP, BestFit, PlotSimilarity и FASTA, которые являются частью пакета программ Accelrys GCG, доступного от Accelrys Software, Inc., Сан-Диего, Калифорния, Соединенные Штаты Америки.
Используемый в данном документе термин "идентичность последовательности" относится к степени, в которой две подвергнутые оптимальному выравниванию полинуклеотидные последовательности или полипептидные последовательности являются инвариантными в окне выравнивания компонентов, например, нуклеотидов или аминокислот. "Идентичность" можно легко рассчитать с помощью известных способов, включающих без ограничения описанные в Computational Molecular Biology (Lesk, A.M., Ed.) Oxford University Press, New York (1988); Biocomputing: Informatics and Genome Projects (Smith, D.W., Ed.) Academic Press, New York (1993); Computer Analysis of Sequence Data, Part I (Griffin, A. M., and Griffin, H.G., Eds.) Humana Press, New Jersey (1994); Sequence Analysis in Molecular Biology (von Heinje, G., Ed.) Academic Press (1987); и Sequence Analysis Primer (Gribskov, M. and Devereux, J., Eds.) Stockton Press, New York (1991).
Используемый в данном документе термин "в значительной степени идентичный" означает, что две нуклеотидные последовательности характеризуются по меньшей мере приблизительно 50%, 60%, 70%, 75%, 80%, 85%, 90% или 95% идентичностью последовательностей. В некоторых вариантах осуществления две нуклеотидные последовательности могут характеризоваться по меньшей мере приблизительно 75%, 80%, 85%, 90%, 95% или 100% идентичностью последовательностей, а также любым диапазоном или значением в нем. В иллюстративных вариантах осуществления две нуклеотидные последовательности могут характеризоваться по меньшей мере приблизительно 95%, 96%, 97%, 98%, 99% или 100% идентичностью последовательностей, а также любым диапазоном или значением в нем.
"Доля идентичности" для выровненных сегментов тестируемой последовательности и эталонной последовательности представляет собой число идентичных компонентов, которые являются общими для двух выровненных последовательностей, разделенное на общее число компонентов в сегменте эталонной последовательности, т.е всей эталонной последовательности или более мелкой определенной части эталонной последовательности. Процентная идентичность последовательностей представляет собой долю идентичности, умноженную на 100. Используемый в данном документе термин "процентная идентичность последовательностей" или "процентная идентичность" относится к процентной доле идентичных нуклеотидов в линейной полинуклеотидной последовательности эталонной ("запрашиваемой") полинуклеотидной молекулы (или ее комплементарной нити) по сравнению с тестируемой ("рассматриваемой") полинуклеотидной молекулой (или ее комплементарной нити), когда две последовательности являются оптимально выровненными (с соответствующими вставками, делециями нуклеотидов или гэпами, в сумме составляющими менее 20 процентов эталонной последовательности на протяжении окна сравнения). В некоторых вариантах осуществления "процентная идентичность" может относиться к процентной доле идентичных аминокислот в аминокислотной последовательности.
Оптимальное выравнивание последовательностей для выравнивания в окне сравнения хорошо известно специалистам в данной области и может осуществляться с помощью инструментов, таких как алгоритм поиска локальной гомологии Смита-Уотермана, алгоритм выравнивания областей гомологии Нидлмана-Вунша, способ поиска подобия Пирсона-Липмана, и необязательно с помощью компьютерных реализаций данных алгоритмов, таких как GAP, BESTFIT, FASTA и TFASTA, доступных в качестве части GCG® Wisconsin Package® (Accelrys Inc., Берлингтон, Массачусетс). Сравнение одной или нескольких полинуклеотидных последовательностей может проводиться с полноразмерной полинуклеотидной последовательностью или ее частью либо с более длинной полинуклеотидной последовательностью. Для целей настоящего изобретения "процентную идентичность" можно также определять с применением BLASTX версии 2.0 для транслируемых нуклеотидных последовательностей и BLASTN версии 2.0 для полинуклеотидных последовательностей.
Процент идентичности последовательностей можно определять с применением программ "Best Fit" или "Gap" из Sequence Analysis Software Package™ (версия 10; Genetics Computer Group, Inc., Мэдисон, Висконсин). В "Gap" используется алгоритм Нидлмана-Вунша (Needleman and Wunsch, J Mol. Biol. 48:443-453, 1970) для нахождения выравнивания двух последовательностей, которое максимально увеличивает число совпадений и сводит к минимуму число гэпов. "BestFit" выполняет оптимальное выравнивание наилучшего сегмента сходства между двумя последовательностями и вставляет гэпы для максимального увеличения числа совпадений с применением алгоритма поиска локальной гомологии Смита-Уотермана (Smith and Waterman, Adv. Appl. Math., 2:482-489, 1981, Smith et al., Nucleic Acids Res. 11:2205-2220, 1983).
Применимые способы определения идентичности последовательностей также раскрыты в Guide to Huge Computers (Martin J. Bishop, ed., Academic Press, San Diego (1994)) и Carillo et al. (AppliedMath 48:1073(1988)). Более конкретно предпочтительные компьютерные программы для определения идентичности последовательностей включают без ограничения программы средства поиска основного локального выравнивания (BLAST), предоставляемые для публичного доступа Национальным центром биотехнологической информации (NCBI) в Национальной библиотеке медицины Национального института здравоохранения, Бетесда, Мэриленд 20894; см. BLAST Manual, Altschul et al., NCBI, NLM, NIH; (Altschul et al., J. Mol. Biol. 215:403-410 (1990)); программы BLAST версии 2.0 или выше позволяют вводить в выравнивания гэпы (делеции и вставки); в случае пептидных последовательностей для определения идентичности последовательностей можно применять BLASTX; а в случае полинуклеотидных последовательностей для определения идентичности последовательностей можно применять BLASTN.
"Гетерозисная группа" содержит набор из генотипов, которые являются эффективными при скрещивании с генотипами из другой гетерозисной группы. Hallauer et al., Corn breeding, in CORN AND CORN IMPROVEMENT p. 463-564 (1998). Инбредные линии классифицируют в гетерозисные группы, и их дополнительно подразделяют в пределах гетерозисной группы на семейства, исходя из нескольких критериев, таких как родословная, связи на основе молекулярных маркеров и характеристики в гибридных комбинациях. Smith et al., Theor. Appl. Gen. 80:833 (1990).
Используемые в данном документе термины "фенотип" или "фенотипический признак" относятся к одному или нескольким признакам организма. Фенотип можно наблюдать невооруженным глазом или с помощью любых других средств для оценки, известных в данной области, например, микроскопии, биохимического анализа и/или электромеханического анализа. В некоторых случаях, фенотип напрямую контролируется одним геном или генетическим локусом, т.е. представляет собой "признак, определяемый одним геном". В других случаях фенотип является результатом действия нескольких генов.
Используемые в данном документе термины "засухоустойчивость" и "засухоустойчивый" относятся к способности растения переносить и/или благополучно развиваться в условиях стресса, вызванного засухой, или в условиях дефицита воды. При применении в отношении идиоплазмы или растения термины относятся к способности растения, которое происходит из такой идиоплазмы или растения, переносить и/или благополучно развиваться в условиях засухи. В общем случае растение или идиоплазма обозначаются как "засухоустойчивые", если они проявляют "повышенную засухоустойчивость".
Используемый в данном документе термин "повышенная засухоустойчивость" относится к улучшению, усилению или повышению в проявлении одного или нескольких фенотипов оптимизации потребления воды по сравнению с одним или несколькими контрольными растениями (например, одним или обоими родителями, или растением, у которого отсутствует маркер, ассоциированный с повышенной засухоустойчивостью). Иллюстративные фенотипы засухоустойчивости включают без ограничений повышенную урожайность в бушелях на акр, урожайность зерна при стандартном процентном содержании влаги (YGSMN), влажность зерна при уборке (GMSTP), вес зерна на делянку (GWTPN), процент восстановления урожайности (PYREC), снижение урожайности (YRED), интервал между пылением и выметыванием пестичных столбиков (ASI) и процент непродуктивности (РВ) (все показатели можно сравнивать в отношении повышения с показателями контрольного растения). Таким образом, растение, которое демонстрирует более высокую YGSMN, чем один или оба ее родителя, когда каждое из них выращивают в условиях стресса, вызванного засухой, проявляет повышенную засухоустойчивость и может обозначаться как "засухоустойчивое".
Используемая в данном документе фраза "абиотический стресс" относится к любому неблагоприятному эффекту в отношении метаболизма, роста, размножения и/или жизнеспособности растения под воздействием абиотических факторов (т.е. водообеспеченности, воздействию тепла, холода и т.д.). Соответственно, абиотический стресс может быть вызван условиями окружающей среды, являющимися субоптимальными для роста, такими как, например, засоленность, водное голодание, водный дефицит, засуха, наводнение, замораживание, низкая или высокая температура (например, охлаждение или чрезмерное нагревание), загрязнение токсичными химическими веществами, токсичность тяжелых металлов, анаэробиоз, недостаточность питательных веществ, избыток питательных веществ, атмосферное загрязнение или УФ-облучение.
Используемая в данном документе фраза "устойчивый к абиотическому стрессу" относится к способности растения переносить абиотический стресс лучше, чем контрольное растение.
Используемые в данном документе термины "дефицит воды" или "засуха" означают период, когда доступная для растения вода не восполняется со скоростью, при которой она потребляется растением. Длительный период дефицита воды в разговорной речи называется засухой. Отсутствие дождя или полива может не приводить немедленно к стрессу, вызванному недостатком воды, если имеется доступный резервуар грунтовых вод для поддержания скорости роста растений. Растения, растущие в почве с достаточным запасом грунтовых вод, могут выживать в течение многих дней без дождя или полива без неблагоприятных воздействий на урожайность. Растения, растущие в сухой почве, вероятно, будут подвергаться неблагоприятным воздействиям при минимальных периодах дефицита воды. Стресс, вызванный тяжелым дефицитом воды, может вызывать увядание и гибель растений; умеренная засуха может снижать урожайность, сдерживать рост или замедлять развитие. Растения могут восстановиться после нескольких периодов стресса, вызванного дефицитом воды, без существенного воздействия на урожайность. Однако дефицит воды во время опыления может уменьшать или снижать урожайность. Таким образом, применимым периодом в жизненном цикле кукурузы, например, для наблюдения за реакцией или устойчивостью к дефициту воды, является поздняя стадия вегетативного роста перед появлением метелки или переходом к развитию репродуктивных органов. Устойчивость к дефициту воды/засухе определяется путем сравнения с контрольными растениями. Например, при воздействии дефицита воды растения по настоящему изобретению могут давать более высокий урожай, чем контрольные растения. В лабораторных условиях и при полевых испытаниях засуху можно имитировать путем подачи растениям по настоящему изобретению и контрольным растениям меньшего количества воды, чем подают контрольным растениям с достаточным уровнем обеспечения водой, и измерения различий в признаках.
Коэффициент использования воды (WUE) представляет собой параметр, часто применяемый для оценки оптимального соотношения между потреблением воды и поглощением CO2/ростом (Kramer, 1983, Water Relations of Plants, Academic Press p. 405). WUE был определен и измерен многочисленными способами. Одним подходом является расчет соотношения сухого веса целого растения и веса воды, потребляемой растением на протяжении всей его жизни (Chu et al., 1992, Oecologia 89:580). Другим вариантом является применение более короткого интервала времени, за который измеряют накопление биомассы и использование воды (Mian et al., 1998, Crop Sci. 38:390). Другой подход представляет собой использование измерений от ограниченных частей растения, например, измерение роста и использования воды только в надземных частях (Nienhuis et al 1994 Amer J Bot 81:943). WUE также был определен как отношение поглощения CO2 к потере воды при испарении из листа или части листа, зачастую измеряемых в течение очень короткого периода времени (например, секунд/минут) (Kramer, 1983, р. 406). Измерение соотношения 13С/12С, фиксируемого в ткани растений, с помощью масс-спектрометра для получения соотношения изотопов также было использовано для оценки WUE у растений, использующих С-3 фотосинтез (Martin et al., 1999, Crop Sci. 1775). Используемый в данном документе термин "коэффициент использования воды" относится к количеству органических веществ, произведенных растением, деленному на количество воды, использованной растением при их производстве, т.е. сухой вес растения по отношению к использованию воды растением. Используемый в данном документе термин "сухой вес" относится ко всем веществам в растении, за исключением воды, и он включает, например, углеводы, белки, масла и минеральные питательные вещества.
Используемый в данном документе термин "ген" относится к единице наследственности, содержащей последовательность ДНК, которая занимает специфическое местоположение в хромосоме и которая содержит генетическую инструкцию для конкретных характеристики или признака, свойственных организму.
Термин "хромосомный интервал" обозначает непрерывный линейный промежуток геномной ДНК, который находится in planta на одной хромосоме. Термин также обозначает любой и все возможные геномные интервалы, определяемые любым из маркеров, изложенных в настоящем изобретении. Генетические элементы, расположенные в одном хромосомном интервале, являются физически сцепленными, и размер хромосомного интервала не является особым образом ограниченным. В некоторых аспектах генетические элементы, расположенные в пределах одного хромосомного интервала, являются физически сцепленными, как правило, расстояние при этом, например, является меньшим или равным 20 млн. о., или в качестве альтернативы является меньшим или равным 10 млн. о. Интервал, описываемый терминальными маркерами, которые определяют конечные точки интервала, будет включать терминальные маркеры и любой маркер, локализованный в пределах такого домена хромосомы, независимо от того, известны или неизвестны такие маркеры в настоящее время. Хотя предполагается, что специалист в данной области может описать дополнительные полиморфные сайты в маркерных локусах и рядом с маркерами, идентифицированными в данном документе, любой маркер в пределах хромосомных интервалов, описанных в данном документе, которые ассоциированы с засухоустойчивостью, находится в пределах объема настоящего заявленного изобретения. Границы хромосомных интервалов содержат маркеры, которые будут сцеплены с геном, генами или локусами, обеспечивающими представляющий интерес признак, т.е. любой маркер, который лежит в пределах рассматриваемого интервала, включая терминальные маркеры, которые определяют границы интервала, можно применять в качестве маркера в отношении засухоустойчивости. Интервалы, описанные в данном документе, охватывают кластеры маркеров, которые косегрерируют с оптимизацией потребления воды при засухоустойчивости. Кластеризация маркеров происходит в относительно небольших доменах на хромосомах, что указывает на присутствие генетического локуса, контролирующего представляющий интерес признак в данных участках хромосомы. Интервал охватывает маркеры, которые картируются в пределах интервала, а также маркеры, которые определяют его концы.
"Локусы количественных признаков" или "локус количественного признака" (QTL) представляют собой домен гена, обеспечивающий фенотип, который можно описать в количественном выражении, и можно установить "фенотипическое значение", которое соответствует количественному значению фенотипического признака. QTL могут функционировать посредством механизма одного гена или с помощью полигенного механизма. Границы хромосомных интервалов наносят так, чтобы они охватывали маркеры, которые будут сцеплены с одним или несколькими QTL. Другими словами, хромосомный интервал наносят таким образом, что любой маркер, который лежит в пределах такого интервала (включая терминальные маркеры, которые определяют границы данного интервала), можно применять в качестве маркеров в отношении засухоустойчивости. Каждый интервал содержит по меньшей мере один QTL, и, кроме того, на самом деле может содержать более одного QTL. Тесное соседство нескольких QTL в одном и том же интервале может затруднять понимание корреляции конкретного маркера с конкретным QTL, поскольку один маркер может демонстрировать сцепление более чем с одним QTL. И наоборот, например, если для двух маркеров, находящихся в тесном соседстве, показана косегрегация с требуемым фенотипическим признаком, иногда непонятно, идентифицирует ли каждый из таких маркеров один и тот же QTL или два разных QTL. Несмотря на это, знание того, сколько QTL находится в конкретном интервале, не является необходимым для проведения или осуществления настоящего изобретения на практике.
Используемая в данном документе фраза "анализ ILLUMINA® GOLDENGATE®" относится к высокопроизводительному анализу генотипирования, реализуемому Illumina Inc. из Сан-Диего, Калифорния, Соединенные Штаты Америки, в котором можно получать SNP-специфические продукты ПЦР. Данный анализ подробно описан на веб-сайте Illumina Inc. и в Fan et al., 2006.
Используемая в данном документе фраза "непосредственно примыкающая", когда она используется при описании молекулы нуклеиновой кислоты, гибридизирующейся с ДНК, содержащей полиморфизм, относится к нуклеиновой кислоте, гибридизирующейся с последовательностью ДНК, которая непосредственно граничит с положением полиморфного нуклеинового основания. Например, молекула нуклеиновой кислоты, которую можно применять в анализе удлинения нити на одно основание, является "непосредственно примыкающей" к полиморфизму.
Используемый в данном документе термин "улучшенный" и его грамматические варианты относятся к растению или его части, потомству или культуре тканей, которые вследствие наличия (или отсутствия) конкретного аллеля, ассоциированного с оптимизацией потребления воды (такого как без ограничения аллели, ассоциированные с оптимизация потребления воды, которые раскрыты в данном документе), характеризуются более высоким или более низким уровнем признака, ассоциированного с оптимизацией потребления воды, в зависимости от того, требуется ли для конкретной цели более высокий или более низкий уровень.
Используемый в данном документе термин "ВСТАВКА/ДЕЛЕЦИЯ" (также записываемый как "вставка/делеция") относится к вставке или делеции в паре нуклеотидных последовательностей, где первая последовательность может рассматриваться как имеющая вставку относительно второй последовательности или вторая последовательность может рассматриваться как имеющая делецию относительно первой
Используемый в данном документе термин "информативный фрагмент" относится к нуклеотидной последовательности, предусматривающей фрагмент большей нуклеотидной последовательности, где данный фрагмент обеспечивает возможность идентификации одного или нескольких аллелей в пределах большей нуклеотидной последовательности. Например, информативный фрагмент нуклеотидной последовательности под SEQ ID NO: 17 содержит фрагмент нуклеотидной последовательности SEQ ID NO: 1 и обеспечивает возможность идентификации одного или нескольких аллелей (например, нуклеотида G в положении 401 из SEQ ID NO: 17), нуклеотидная последовательность под SEQ ID NO: 18 содержит фрагмент нуклеотидной последовательности SEQ ID NO: 2 и обеспечивает возможность идентификации одного или нескольких аллелей (например, нуклеотида G в положении 401 из SEQ ID NO: 18), нуклеотидная последовательность под SEQ ID NO: 19 содержит фрагмент нуклеотидной последовательности SEQ ID NO: 3 и обеспечивает возможность идентификации одного или нескольких аллелей (например, нуклеотида А в положении 401 из SEQ ID NO: 19), нуклеотидная последовательность под SEQ ID NO: 20 содержит фрагмент нуклеотидной последовательности SEQ ID NO: 4 и обеспечивает возможность идентификации одного или нескольких аллелей (например, нуклеотида А в положении 401 из SEQ ID NO: 20), нуклеотидная последовательность под SEQ ID NO: 21 содержит фрагмент нуклеотидной последовательности SEQ ID NO: 5 и обеспечивает возможность идентификации одного или нескольких аллелей (например, нуклеотида G в положении 401 из SEQ ID NO: 21), нуклеотидная последовательность под SEQ ID NO: 22 содержит фрагмент нуклеотидной последовательности SEQ ID NO: 6 и обеспечивает возможность идентификации одного или нескольких аллелей (например, нуклеотида С в положении 401 из SEQ ID NO: 22), нуклеотидная последовательность под SEQ ID NO: 23 содержит фрагмент нуклеотидной последовательности SEQ ID NO: 7 и обеспечивает возможность идентификации одного или нескольких аллелей (например, нуклеотида А в положении 401 из SEQ ID NO: 23), и нуклеотидная последовательность под SEQ ID NO: 24 содержит фрагмент нуклеотидной последовательности SEQ ID NO: 8 и обеспечивает возможность идентификации одного или нескольких аллелей (например, нуклеотида G в положении 401 из SEQ ID NO: 24).
Используемая в данном документе фраза "проверочное положение" относится к физическому положению на твердой подложке, которое можно запрашивать для получения данных генотипирования в отношении одного или нескольких заранее заданных геномных полиморфизмов.
Используемый в данном документе термин "полиморфизм" относится к вариации нуклеотидной последовательности в локусе, в котором указанная вариация является слишком распространенной, чтобы быть вызванной всего лишь спонтанной мутацией. Полиморфизм должен иметь частоту в популяции, составляющую по меньшей мере приблизительно 1%. Полиморфизм может представлять собой однонуклеотидный полиморфизм (SNP) или инсерционно-делеционный полиморфизм, также называемый в данном документе "вставкой/делецией." Дополнительно изменчивость может наблюдаться в транскрипционном профиле или паттерне метилирования. Полиморфный сайт или сайты нуклеотидной последовательности можно определить путем сравнения нуклеотидных последовательностей в одном или нескольких локусах в двух или более элементах идиоплазмы.
Используемая в данном документе фраза "рекомбинация" относится к обмену фрагментами ДНК между двумя молекулами ДНК или хроматидами парных хромосом ("кроссинговеру") в участке сходных или идентичных нуклеотидных последовательностей. "Событие рекомбинации" в данном документе понимают как обозначающее мейотический кроссинговер.
Используемый в данном документе термин "растение" может относиться к целому растению, любой его части или культуре клеток или тканей, полученным из растения. Таким образом, термин "растение" может относиться к целому растению, части растения или органу растения (например, листьям, стеблям, корням и т.д.), растительной ткани, семени и/или растительной клетке. Растительная клетка представляет собой клетку растения, взятую из растения или полученную посредством культивирования из клетки, взятой из растения.
Используемый в данном документе термин "маис" относится к растению Zea mays L. ssp. mays и также известному как "кукуруза".
Используемый в данном документе термин "растение маиса" включает целые растения маиса, клетки растения маиса, протопласт растения маиса, культуры клеток растения маиса или тканей маиса, из которых можно регенерировать растения маиса, каллюсы растения маиса и клетки растения маиса, которые являются интактными в растениях маиса или частях растений маиса, таких как семена маиса, початки маиса, цветки маиса, семядоли маиса, листья маиса, стебли маиса, почки маиса, корни маиса, кончики корней маиса и т.п.
Используемая в данном документе фраза "нативный признак" относится к любому существующему моногенному или олигогенному признаку в идиоплазме определенной сельскохозяйственной культуры. В случае идентификации за счет молекулярного(-ых) маркера(-ов) полученную информацию можно применять для улучшения идиоплазмы посредством селекции с помощью маркера по признакам, ассоциированным с оптимизацией потребления воды, раскрытым в данном документе.
"Не встречающийся в природе сорт маиса" представляет собой сорт маиса, который в естественном состоянии не встречается в природе. "Не встречающийся в природе сорт маиса" может быть получен с помощью любого способа, известного в данной области, включая без ограничений трансформацию растения или идиоплазмы маиса, трансфекцию растения или идиоплазмы маиса и скрещивание встречающегося в природе сорта маиса с не встречающимся в природе сортом маиса, посредством редактирования генома (например, CRISPR или TALEN) или за счет создания селекционных пакетов из требуемых аллелей, не присутствующих в природе. В некоторых вариантах осуществления "не встречающийся в природе сорт маиса" может содержать одну из множества гетерологичных нуклеотидных последовательностей. В некоторых вариантах осуществления "не встречающийся в природе сорт маиса" может содержать одну или несколько не встречающихся в природе копий встречающейся в природе нуклеотидной последовательности (т.е. лишние копии гена, который естественным образом встречается в маисе).
Гетерозисная группа "non-Stiff Stalk" представляет собой основную гетерозисную группу в северных регионах выращивания кукурузы в США и Канаде. Она может также обозначаться как гетерозисная группа "Lancaster" или "Lancaster Sure Crop".
Гетерозисная группа "Stiff Stalk" представляет собой основную гетерозисную группу в северных регионах выращивания кукурузы в США и Канаде. Она может также обозначаться как гетерозисная группа "Iowa Stiff Stalk Synthetic" или "BSSS".
Используемый в данном документе термин "процент непродуктивности" (РВ) относится к процентной доле растений в рассматриваемой области (например, делянке) с отсутствием зерна. Как правило, его выражают в виде процентной доли растений на делянку, и его можно рассчитать как:

Используемый в данном документе термин "процент восстановления урожайности" (PYREC) относится к влиянию, которое аллель и/или комбинация аллелей оказывает на урожайность растения, выращиваемого в условиях стресса, вызванного засухой, по сравнению с урожайностью растения, которое является генетически идентичным за исключением того, что у него отсутствует данный аллель и/или комбинация аллелей. PYREC рассчитывают следующим образом:

В качестве примера, а не ограничения, если контрольное растение приносит урожай 200 бушелей в условиях обильного полива, но приносит урожай только 100 бушелей в условиях стресса, вызванного засухой, то его процентная доля потери урожая будет рассчитана как 50%. Если в иных отношениях генетически идентичный гибрид, который содержит представляющий интерес аллель(-и), приносит урожай 125 бушелей в условиях стресса, вызванного засухой, и 200 бушелей в условиях обильного полива, то процентная доля потери урожая будет рассчитана как 37,5%, a PYREC будет рассчитан как 25% [1,00-(200-125)/(200-100)×100)].
Используемая в данном документе фраза "урожайность зерна при достаточном наличии воды" относится к урожаю из области, которая получала достаточный полив, чтобы предотвратить недостаток воды у растений на протяжении их цикла роста. В некоторых вариантах осуществления этот признак выражают в бушелях на акр.
Используемая в данном документе фраза "снижение урожайности у гибридных линий" относится к рассчитанному признаку, полученному на основании испытания по изучению урожайности у гибридных линий, выращиваемых в условиях стресса и в условиях отсутствия стресса. Для рассматриваемой гибридной линии это составляет:

В некоторых вариантах осуществления этот признак выражают в виде процента бушелей на акр.
Используемая в данном документе фраза "снижение урожайности у инбредных линий" относится к рассчитанному признаку, полученному на основании испытания по изучению урожайности у инбредных линий, выращиваемых в условиях стресса и в условиях отсутствия стресса. Для рассматриваемой инбредной линии это составляет:

В некоторых вариантах осуществления этот признак выражают в виде процента бушелей на акр.
Используемые в данном документе термины "нуклеотидная последовательность", "полинуклеотид," "последовательность нуклеиновой кислоты", "молекула нуклеиновой кислоты" и "фрагмент нуклеиновой кислоты" относятся к полимеру РНК или ДНК, которая является одно- или двухнитевой, необязательно содержащей синтетические, не встречающиеся в природе и/или измененные нуклеиновые основания. "Нуклеотид" представляет собой мономерную единицу, из которой сконструированы полимеры ДНК или РНК и которая состоит из пуринового или пиримидинового основания, пентозы и группы фосфорной кислоты. Нуклеотиды (обычно находящиеся в своей форме 5'-монофосфата) называют их однобуквенным обозначением следующим образом: "А" в случае аденилата или дезоксиаденилата (соответственно, в случае РНК или ДНК), "С" в случае цитидилата или дезоксицитидилата, "G" в случае гуанилата или дезоксигуанилата, "U" в случае уридилата, "Т" в случае дезокситимидилата, "R" в случае пуринов (А или G), "Y" в случае пиримидинов (С или Т), "K" в случае G или Т, "Н" в случае А, или С, или Т, "I" в случае инозина и "N" в случае любого нуклеотида.
Используемый в данном документе термин "часть растения" включает без ограничения зародыши, пыльцу, семена, листья, цветки (включая без ограничения пыльники, семязачатки и т.п.), плоды, стебли или ветви, корни, кончики корней, клетки, включая клетки, которые являются интактными в растениях и/или частях растений, протопласты, культуры клеток и тканей растений, каллюсы растений, скопления клеток растений и т.п. Таким образом, часть растения включает культуру тканей сои, из которой можно регенерировать растения сои. Кроме того, используемый в данном документе термин "клетка растения" относится к структурной и физиологической единице растения, которая содержит клеточную стенку, а также может относиться к протопласту. Клетка растения по настоящему изобретению может находиться в виде отдельной выделенной клетки, или может представлять собой культивируемую клетку, или может представлять собой часть более высокоорганизованной единицы, такой как, например, ткань растения или орган растения.
Используемый в данном документе термин "популяция" относится к генетически гетерогенной совокупности растений, имеющих общее генетическое происхождение.
Используемые в данном документе термины "потомство", "растение-потомок" и/или "потомок" относятся к растению, полученному в результате вегетативного или полового размножения одного или нескольких родительских растений. Растение-потомок может быть получено путем клонирования или самоопыления одного родительского растения или путем скрещивания двух родительских растений, и оно включает продукты самоопыления, а также F1 или F2 или более далекие поколения. F1 представляет собой потомство первого поколения, полученное от родительских особей, по меньшей мере одну из которых впервые используют в качестве донора признака, тогда как потомство второго поколения (F2) или последующих поколений (F3, F4 и т.п.) представляет собой образцы, полученные в результате самоопыления или скрещивания F1, F2 и т.п. Таким образом, F1 может представлять собой (и в некоторых вариантах осуществления представляет собой) гибрид, полученный в результате скрещивания двух родительских особей из чистых линий (фраза "чистая линия" относится к особи, гомозиготной по одному или нескольким признаками), тогда как F2 может представлять собой потомство, полученное в результате гомоклинного опыления гибридов F1.
Используемый в данном документе термин "эталонная последовательность" относится к определенной нуклеотидной последовательности, применяемой в качестве основы для сравнения нуклеотидных последовательностей (например, хромосома 1 или хромосома 3 культивара В73 Zea mays). Эталонную последовательность для маркера, например, можно получить путем генотипирования ряда линий в представляющем интерес локусе или локусах, выравнивания нуклеотидных последовательностей в программе для выравнивания последовательностей, а затем получения консенсусной последовательности выравнивания. Следовательно, с помощью эталонной последовательности идентифицируют полиморфизмы в аллелях в локусе. Эталонная последовательность может не быть копией действительной последовательности нуклеиновой кислоты из любого конкретного организма; однако она пригодна для конструирования праймеров и зондов для выявления действительных полиморфизмов в локусе или локусах.
Используемый в данном документе термин "выделенная" относится к нуклеотидной последовательности (например, генетическому маркеру), которая не содержит последовательности, которые в норме фланкируют одну или обе стороны данной нуклеотидной последовательности в геноме растения. Соответственно, фраза "выделенный и очищенный генетический маркер, ассоциированный с признаком оптимизации потребления воды у Zea mays", может обозначать, например, молекулу рекомбинантной ДНК при условии, что одна из последовательностей нуклеиновой кислоты, которая в норме обнаруживается, как фланкирующая такую молекулу рекомбинантной ДНК во встречающемся в природе геноме, является удаленной или отсутствует. Таким образом, выделенные нуклеиновые кислоты включают без ограничений рекомбинантную ДНК, которая существует в виде отдельной молекулы (включая без ограничений фрагменты геномной ДНК, полученной с помощью ПЦР или обработки рестрикционными эндонуклеазами) с отсутствием каких-либо фланкирующих последовательностей, а также рекомбинантную ДНК, которая встроена в вектор, автономно реплицирующуюся плазмиду или в геномную ДНК растения в качестве части молекулы гибридной или слитой нуклеиновой кислоты.
Используемая в данном документе фраза "анализ TAQMAN®" относится к выявлению последовательности в режиме реального времени с применением ПЦР на основе анализа TAQMAN®, реализуемого Applied Biosystems, Inc. из Фостер-Сити, Калифорния, Соединенные Штаты Америки. В случае идентифицированного маркера можно разработать анализ TAQMAN® для применения в селекционной программе.
Используемый в данном документе термин "тестер" относится к линии, применяемой в тест-кроссе с одним или несколькими другими линиями, где тестер и тестируемая линия являются генетически разнородными. Тестер может представлять собой изогенную линию по отношению к скрещиваемой линии.
Используемый в данном документе термин "признак" относится к представляющему интерес фенотипу, гену, вносящему вклад в представляющий интерес фенотип, а также к последовательности нуклеиновой кислоты, ассоциированной с геном, вносящим вклад в представляющий интерес фенотип. Например, "признак оптимизации потребления воды" относится к фенотипу оптимизации потребления воды, а также к гену, вносящему вклад в фенотип оптимизации потребления воды, и последовательности нуклеиновой кислоты (например, SNP или другому маркеру), который ассоциирован с фенотипом оптимизации потребления воды.
Используемый в данном документе термин "трансген" относится к молекуле нуклеиновой кислоты, введенной в организм или его предков с помощью некоторой формы методики искусственного переноса. Таким образом, с помощью методики искусственного переноса создают "трансгенный организм" или "трансгенную клетку". Следует понимать, что методика искусственного переноса может осуществляться в организме-предке (или в его клетке и/или клетке, из которой может развиваться организм-предок), и при этом любая особь-потомок, которая имеет искусственно перенесенную молекулу нуклеиновой кислоты или ее фрагмент, все еще считается трансгенной, даже если в результате одного или нескольких естественных и/или принудительных скрещиваний искусственно перенесенная молекула нуклеиновой кислоты присутствует в особи-потомке.
«Неблагоприятный аллель» представляет собой маркерный аллель, который сегрегирует с неблагоприятным фенотипом растения, обеспечивая тем самым преимущество идентификации растений, которые можно удалять из селекционной программы или высаживания.
Используемый в данном документе термин «оптимизация потребления воды» относится к любому параметру растения, его частей или его структуры, который может быть измерен и/или количественно определен, чтобы оценить степень или скорость роста и развитие растений в условиях достаточной водообеспеченности по сравнению с условиями водообеспеченности ниже оптимальных (например, засухи). В связи с этим "признак оптимизации потребления воды" представляет собой любой признак, который, как может быть показано, влияет на урожайность растения при различных наборах условий роста, относящихся к водообеспеченности. Используемая в данном документе фраза "оптимизация потребления воды" относится к любому параметру растения, его частей или его структуры, который может быть измерен и/или количественно определен, чтобы оценить степень или скорость роста и развития растений при различных условиях водообеспеченности. (Например, все маркерные аллели, идентифицированные в таблицах 1-7, или их тесно сцепленные маркеры можно использовать для идентификации, отбора или получения растений маиса, характеризующихся повышенной оптимизацией потребления воды). Аналогично "оптимизация потребления воды" может считаться "фенотипом", который, как используется в данном документе, относится к выявляемой, наблюдаемой и/или измеряемой характеристике клетки или организма. В некоторых вариантах осуществления фенотип основан по меньшей мере частично на генетической структуре клетки или организма (называемой в данном документе "генотипом" клетки или организма). Иллюстративные фенотипы оптимизации потребления воды представляют собой урожайность зерна при стандартном процентном содержании влаги (YGSMN), влажность зерна при уборке (GMSTP), вес зерна на делянку (GWTPN) и процент восстановления урожайности (PYREC). Следует отметить, что используемый в данном документе термин "фенотип" учитывает то, как окружающая среда (например, условия окружающей среды) может воздействовать на оптимизацию потребления воды, вследствие чего эффект оптимизации потребления воды является реальным и воспроизводимым. Используемый в данном документе термин "снижение урожайности" (YD) относится к степени, до которой снижается урожайность растений, выращиваемых в условиях стресса. YD рассчитывают следующим образом:

Генетические локусы, коррелирующие с конкретными фенотипами, такими как засухоустойчивость, можно картировать в геноме организма. Посредством идентификации маркера или кластера маркеров, которые косегрегируют с представляющим интерес признаком, селекционер способен быстро отобрать требуемый фенотип путем отбора по надлежащему маркеру (способ, называемый отбором с помощью маркеров или MAS). Такие маркеры также могут применяться селекционерами для разработки генотипов in silico и для осуществления полногеномного отбора на практике.
В настоящем изобретении предусмотрены хромосомные интервалы, QTL, локусы и гены, ассоциированные с улучшенной засухоустойчивостью растений (например, маиса) и/или улучшенной/повышенной урожайностью растения (например, маиса). Выявление этих маркеров и/или других сцепленных маркеров можно применять для идентификации, отбора и/или получения растений маиса, характеризующихся повышенной засухоустойчивостью, и/или для исключения из селекционных программ или высаживания растений маиса, которые не характеризуются повышенной засухоустойчивостью.
Молекулярные маркеры применяют для визуализации различий в последовательностях нуклеиновой кислоты. Данная визуализация может быть обусловлена методиками ДНК-ДНК гибридизации после расщепления рестрикционным ферментом (например, RFLP) и/или обусловлена методиками с применением полимеразной цепной реакции (например, SNP, STS, SSR/микросателлиты, AFLP и т.п.). В некоторых вариантах осуществления все отличия между двумя родительскими генотипами сегрегируют в картирующей популяции на основе скрещивания данных родительских генотипов. Сегрегацию разных маркеров можно сравнивать, и можно рассчитывать значения частоты рекомбинации. Способы картирования маркеров в растениях раскрыты, например, в Glick & Thompson (1993) Methods in Plant Molecular Biology and Biotechnology, CRC Press, Boca Raton, Florida, United States of America; Zietkiewicz et al. (1994) Genomics 20:176-183.
В таблицах 1-8 приведены названия участков генома Zea maize (т.е. хромосомных интервалов, генов, QTL, аллелей или локусов), физические генетические местоположения каждого маркера на соответствующей хромосоме или группе сцепления маиса и целевой аллель(-и), которые ассоциированы с повышенной засухоустойчивостью, оптимизацией потребления воды и/или урожайностью маиса либо в условиях засухи, либо в условиях, отличных от засухи. Маркеры по настоящему изобретению описаны в данном документе со ссылкой на положения маркерных локусов, картированных по физическим местоположениям, как они сообщаются в публично доступной сборке последовательности В73 RefGen_v2 от Arizona Genomics Institute. Физическую последовательность генома маиса можно найти на интернет-ресурсах: maizeGDB (maizegdb.org/assembly) или Gramene на (gramene.org).
Таким образом, в некоторых вариантах осуществления настоящего изобретения маркерные аллели, хромосомные интервалы и/или QTL, ассоциированные с повышенной засухоустойчивостью или повышенной урожайностью в условиях засухи или в условиях, отличных от засухи, изложены в таблицах 1-7.
В некоторых вариантах осуществления настоящего изобретения маркерный(-е) аллель(-и) и их тесно сцепленные маркеры, ассоциированные с повышенной засухоустойчивостью, изложенные в таблицах 1-7, могут быть расположены в хромосомном интервале, включающем без ограничений (а) хромосомный интервал на хромосоме 1, определяемый от положения пары оснований (п. о.) 272937470 до положения пары оснований (п. о.) 272938270 включительно (PZE01271951242); (b) хромосомный интервал на хромосоме 2, определяемый от положения пары оснований (п. о.) 12023306 до положения пары оснований (п. о.) 12024104 включительно (PZE0211924330); (с) хромосомный интервал на хромосоме 3, определяемый от положения пары оснований (п. о.) 225037202 до положения пары оснований (п. о.) 225038002 включительно (PZE03223368820); (d) хромосомный интервал на хромосоме 3, определяемый от положения пары оснований (п. о.) 225340531 до положения пары оснований (п. о.) 225341331 включительно (PZE03223703236); (е) хромосомный интервал на хромосоме 5, определяемый от положения пары оснований (п. о.) 159120801 до положения пары оснований (п. о.) 159121601 включительно (PZE05158466685); (f) хромосомный интервал на хромосоме 9, определяемый от положения пары оснований (п. о.) 12104536 до положения пары оснований (п. о.) 12105336 включительно (PZE0911973339); (g) хромосомный интервал на хромосоме 9, определяемый от положения пары оснований (п. о.) 225343590 до положения пары оснований (п. о.) 225340433 включительно (S_l8791654); (h) хромосомный интервал на хромосоме 10, определяемый от положения пары оснований (п. о.) 14764415 до положения пары оснований (п. о.) 14765098 включительно (S_20808011), или любую их комбинацию. Как будет понятно специалисту в данной области, дополнительные хромосомные интервалы могут определяться маркерами SNP, предусмотренными в данном документе в таблице 1. Кроме того, маркеры SNP в пределах хромосомных интервалов (а) - (h), отличные от тех, которые предусмотрены в таблице 1, могут быть получены с помощью способов, хорошо известных в данной области.
В настоящем изобретении дополнительно предусмотрено, что выявление молекулярного маркера может предусматривать применение зонда на основе нуклеиновой кислоты, имеющего последовательность нуклеиновых оснований, которая в значительной степени комплементарна последовательности нуклеиновой кислоты, определяющей молекулярный маркер, и при этом зонд на основе нуклеиновой кислоты специфически гибридизируется в жестких условиях с последовательностью нуклеиновой кислоты, определяющей молекулярный маркер. Подходящий зонд на основе нуклеиновой кислоты, например, может представлять собой одну нить из продукта амплификации, соответствующего маркеру. В некоторых вариантах осуществления выявление маркера предназначено для определения того, присутствует или отсутствует конкретный аллель SNP в конкретном растении.
Кроме того, способы по настоящему изобретению включают выявление амплифицированного фрагмента ДНК, ассоциированного с присутствием конкретного аллеля SNP. В некоторых вариантах осуществления амплифицированный фрагмент, ассоциированный с конкретным аллелем SNP, имеет предсказанную длину и/или последовательность нуклеиновой кислоты, и выявление амплифицированного фрагмента ДНК, имеющего предсказанную длину или предсказанную последовательность нуклеиновой кислоты, проводят таким образом, чтобы амплифицированный фрагмент ДНК фрагмент имел длину, которая соответствует (плюс-минус несколько оснований; например, длину, которая меньше или больше на одно, два или три основания) ожидаемой длине, исходя из аналогичной реакции с теми же праймерами с ДНК из растения, в котором маркер был впервые выявлен, или последовательность нуклеиновой кислоты, которая соответствует (например, характеризуется гомологией, составляющей по меньшей мере приблизительно 80%, 90%, 95%, 96%, 97%, 98%, 99% или больше) ожидаемой последовательности, исходя из последовательности маркера, ассоциированного с таким SNP в растении, в котором маркер был впервые выявлен.
Выявление амплифицированного фрагмента ДНК, имеющего предсказанную длину или предсказанную последовательность нуклеиновой кислоты, можно проводить с помощью ряда методик, включая без ограничения стандартные методики гель-электрофореза, или с применением автоматизированного ДНК-секвенатора. Такие способы выявления амплифицированного фрагмента ДНК не описаны в данном документе подробно, поскольку они хорошо известны обычным специалистам в данной области.
Как показано в таблицах 1-8, маркеры SNP по настоящему изобретению являются ассоциированными с повышенной засухоустойчивостью и/или повышенной урожайностью либо в условиях засухи, либо в условиях, отличных от засухи. В некоторых вариантах осуществления, как описано в данном документе, один маркер или комбинацию маркеров можно применять для выявления присутствия засухоустойчивого растения маиса или растений маиса, характеризующихся повышенной урожайностью в условиях, отличных от засухи, по сравнению с контрольным растением. В некоторых вариантах осуществления маркер может быть расположен в пределах хромосомного интервала (QTL) или присутствовать в геноме растения в качестве гаплотипа, определяемого в данном документе (например, любого из хромосомных интервалов 1, 2, 3, 4, 5, 6 или 7, определяемых в данном документе).
II. Молекулярные маркеры, локусы, ассоциированные с оптимизацией потребления воды, и композиции для анализа последовательностей нуклеиновой кислоты
Молекулярные маркеры применяют для визуализации различий в последовательностях нуклеиновой кислоты. Данная визуализация может быть обусловлена методиками ДНК-ДНК гибридизации после расщепления рестрикционным ферментом (например, RFLP) и/или обусловлена методиками с применением полимеразной цепной реакции (например, STS, SSR/микросателлиты, AFLP и т.п.). В некоторых вариантах осуществления все отличия между двумя родительскими генотипами сегрегируют в картирующей популяции на основе скрещивания данных родительских генотипов. Сегрегацию разных маркеров можно сравнивать, и можно рассчитывать значения частоты рекомбинации. Способы картирования маркеров в растениях раскрыты, например, в Glick & Thompson, 1993; Zietkiewicz et al., 1994. Частоты рекомбинации молекулярных маркеров на разных хромосомах обычно составляют 50%. Частота рекомбинации между молекулярными маркерами, расположенными на одной и той же хромосоме, обычно зависит от расстояния между маркерами. Низкая частота рекомбинации, как правило, соответствует небольшому генетическому расстоянию между маркерами на хромосоме. Сравнение всех частот рекомбинации приводит к наиболее логичному порядку расположения молекулярных маркеров на хромосомах. Данный наиболее логичный порядок расположения может быть изображен на карте сцепления (Paterson, 1996). Группа смежных или непрерывных маркеров на карте сцепления, ассоциированных с повышенной оптимизацией потребления воды, может определять положение MTL, ассоциированного с повышенной оптимизацией потребления воды. Генетические локусы, коррелирующие с конкретными фенотипами, такими как засухоустойчивость, можно картировать в геноме организма. Посредством идентификации маркера или кластера маркеров, которые косегрегируют с представляющим интерес признаком, селекционер способен быстро отобрать требуемый фенотип путем отбора по надлежащему маркеру (способ, называемый отбором с помощью маркеров или MAS). Такие маркеры также могут применяться селекционерами для конструирования генотипов in silico и для осуществления полногеномного отбора на практике.
В некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, предусмотрены маркеры, ассоциированные с повышенной засухоустойчивостью/оптимизацией потребления воды (например, маркеры, показанные в таблицах 1-7). Выявление данных маркеров и/или других сцепленных маркеров можно использовать для идентификации, отбора и/или получения засухоустойчивых растений и/или для устранения растений, которые не являются засухоустойчивыми, из программ селекции или высаживания.
В некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, последовательности ДНК в пределах 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20 или 25 сМ от маркера из таблиц 1-7 демонстрируют частоту генетической рекомбинации с маркером из объектов, раскрытых в настоящем изобретении, составляющую меньше приблизительно 25%, 20%, 15%, 10%, 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2% или 1%. В некоторых вариантах осуществления идиоплазма представляет собой линию или сорт Zea mays.
Также предусмотрены фрагменты ДНК, ассоциированные с присутствием признака, аллелей и/или гаплотипов, ассоциированных с оптимизацией потребления воды, включая без ограничения SEQ ID NO: 17-24. В некоторых вариантах осуществления фрагменты ДНК, ассоциированные с присутствием признака, ассоциированного с оптимизацией потребления воды, имеют предсказанную длину и/или последовательность нуклеиновой кислоты, а выявление фрагмента ДНК, имеющего предсказанную длину и/или предсказанную последовательность нуклеиновой кислоты проводят с тем, чтобы амплифицированный фрагмент ДНК имел длину, которая соответствует (плюс-минус несколько оснований; например, длину, которая больше или меньше на один, два или три основания) предсказанной длине. В некоторых вариантах осуществления фрагмент ДНК представляет собой амплифицированный фрагмент, и амплифицированный фрагмент имеет предсказанную длину и/или последовательность нуклеиновой кислоты, такую как у амплифицированного фрагмента, полученного с помощью аналогичной реакции с теми же праймерами с помощью ДНК из растения, в котором данный маркер был выявлен впервые, или последовательность нуклеиновой кислоты, которая соответствует (т.е. характеризуется идентичностью нуклеотидной последовательности, составляющей более 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99%) ожидаемой последовательности, исходя из последовательности маркера, ассоциированного с таким признаком, ассоциированным с оптимизацией потребления воды, в растении, в котором маркер был выявлен впервые. После просмотра настоящего изобретения средний специалист в данной области поймет, что маркеры, которые отсутствуют в растениях, хотя они присутствовали по меньшей мере в одном родительском растении (так называемые транс-маркеры), также могут быть применимы в анализах для выявления требуемого признака в растении-потомке, хотя тестирование в отношении отсутствия маркера для выявления присутствия специфического признака не является оптимальным. Выявление амплифицированного фрагмента ДНК, имеющего предсказанную длину или предсказанную последовательность нуклеиновой кислоты, можно проводить с помощью ряда методик, включая без ограничения методики стандартного гель-электрофореза, и/или с помощью автоматизированного ДНК-секвенатора. Поскольку данные способы хорошо известны специалисту в данной области, в данном документе они не описываются подробно.
В некоторых вариантах осуществления праймер (в некоторых вариантах осуществления праймер для удлинения, а в некоторых вариантах осуществления праймер для амплификации) является однонитевым для максимальной эффективности удлинения и/или амплификации. В некоторых вариантах осуществления праймер представляет собой олигодезоксирибонуклеотид. Праймер, как правило, является достаточно длинным, чтобы служить затравкой для синтеза продуктов удлинения и/или амплификации в присутствии средства для полимеризации. Значения минимальной длины праймера могут зависеть от многих факторов, в том числе без ограничения от температуры и состава (содержания А/Т по сравнению с G/C) праймера.
В контексте праймера для амплификации данные праймеры, как правило, предусмотрены в виде одного или нескольких наборов из двунаправленных праймеров, которые включают один или несколько прямых и один или несколько обратных праймеров, обычно применяемых в области амплификации ДНК, как, например, при ПЦР-амплификации. Соответственно, будет понятно, что используемый в данном документе термин "праймер" может относиться к более чем одному праймеру, в частности, в случае, когда существует некоторая неясность в информации о концевой последовательности(-ях) целевой области, подлежащей амплификации. Следовательно, "праймер" может включать совокупность праймерных олигонуклеотидов, содержащих последовательности, представляющие возможные вариации в последовательности, или он содержит нуклеотиды, обеспечивающие возможность обычного спаривания оснований. Праймеры можно получить любым подходящим способом. Способы получения олигонуклеотидов с конкретной последовательностью известны из уровня техники и включают, например, клонирование и рестрикцию соответствующих последовательностей, а также прямой химический синтез. Способы химического синтеза могут включать, например, фосфодиэфирный или фосфотриэфирный способ, диэтилфосфорамидатный способ и способ с использованием твердых подложек, раскрытый в патенте США №4458068.
При необходимости праймеры можно пометить путем включения в их состав выявляемых фрагментов, например, спектроскопических, флуоресцентных, фотохимических, биохимических, иммунохимических или химических фрагментов.
Зависимое от матрицы удлинение олигонуклеотидного праймера катализируется средством для полимеризации в присутствии достаточных количеств четырех дезоксирибонуклеотидтрифосфатов (dATP, dGTP, dCTP и dTTP; т.e. dNTP) или аналогов в реакционной среде, которая содержит подходящие соли, катионы металлов, буферизующую систему для рН. Подходящие средства для полимеризации представляют собой ферменты, которые, как известно, катализируют зависимый от праймера и матрицы синтез ДНК. Известные ДНК-полимеразы включают, например, ДНК-полимеразу из Е. coli или ее фрагмент Кленова, Т4 ДНК-полимеразу и Taq ДНК-полимеразу, а также их различные модифицированные версии. Условия реакции для катализа синтеза ДНК с помощью данных ДНК-полимераз известны из уровня техники. Продуктами синтеза являются дуплексные молекулы, состоящие из нитей-матриц и нитей удлинения праймеров, которые содержат целевую последовательность. Эти продукты, в свою очередь, могут служить в качестве матрицы для следующего раунда репликации. Во втором раунде репликации нить удлинения праймера из первого раунда отжигается с комплементарным ей праймером; при этом синтез дает "короткий" продукт, который связан на обоих 5'- и 3'-концах с праймерными последовательностями или комплементарными им последовательностями. В результате повторяющихся циклов денатурации, отжига и удлинения праймеров может происходить экспоненциальное накопление целевого участка, определяемого праймерами. Для достижения требуемого количества полинуклеотида, содержащего целевой участок нуклеиновой кислоты, проводят достаточное число циклов. Требуемое количество может варьировать, и оно определяется функцией, для которой служит полинуклеотидный продукт.
Способ ПЦР хорошо описан в руководствах и известен специалисту в данной области. После амплификации путем ПЦР целевые полинуклеотиды можно выявлять посредством гибридизации с полинуклеотидным зондом, который образует стабильный гибрид с целевыми полинуклеотидами целевой последовательности в условиях гибридизации и отмывки от жестких до умеренно жестких. Если ожидается, что зонды будут фактически полностью комплементарны (т.е. на приблизительно 99% или больше) целевой последовательности, можно применять жесткие условия. Если ожидается некоторое несовпадение, например, если ожидается наличие вариантов штаммов, вследствие чего зонд не будет полностью комплементарен, то жесткость гибридизации можно уменьшить. В некоторых вариантах осуществления условия выбирают для исключения неспецифического/случайного связывания. Условия, которые влияют на гибридизацию, и которые выбирают для предупреждения неспецифического связывания, известны из уровня техники и описаны, например, в Sambrook & Russell, 2001. В целом, более низкая концентрация солей и более высокая температура повышают жесткость условий гибридизации.
Для выявления присутствия на одной хромосоме в растении двух аллелей, ассоциированных с оптимизацией потребления воды, можно также использоваться способы окрашивания хромосом. В таких способах по меньшей мере первый аллель, ассоциированный с оптимизацией потребления воды, и по меньшей мере второй аллель, ассоциированный с оптимизацией потребления воды, можно выявлять в одной и той же хромосоме с помощью in situ гибридизации или in situ ПЦР-методик. Удобнее всего тот факт, что два аллеля, ассоциированные с оптимизацией потребления воды, присутствуют на одной хромосоме можно подтверждать путем определения того, что они находятся в фазе сцепления: т.е., что данные признаки показывают сниженную сегрегацию по сравнению с генами, находящимися на отдельных хромосомах.
Идентифицированные в данном документе аллели, ассоциированные с оптимизацией потребления воды, расположены на нескольких разных хромосомах или группах сцепления, и их местоположения можно охарактеризовать с помощью целого ряда иных произвольных маркеров. В исследованиях по настоящему изобретению применяли однонуклеотидные полиморфизмы (SNP), хотя также можно было бы применять маркеры полиморфизма длин рестрикционных фрагментов (RFLP), маркеры полиморфизма длин амплифицированных фрагментов (AFLP), микросателлитные маркеры (например, SSR), маркеры инсерционных мутаций, маркеры амплифицированного участка, охарактеризованного последовательностью (SCAR), маркеры рестрикционного полиморфизма амплифицированных последовательностей (CAPS), изоферментные маркеры, технологии на основе микрочипов, анализы TAQMAN®, анализ ILLUMINA® GOLDENGATE®, технологии секвенирования нуклеиновых кислот или комбинации этих маркеров, и их в действительности можно применять.
Обычно предоставление полной информации о последовательности для аллеля и/или гаплотипа, ассоциированных с оптимизированным потреблением воды, является необязательным, поскольку способ, посредством которого аллель и/или гаплотип, ассоциированные с оптимизацией потребления воды, впервые выявлен - за счет наблюдаемой корреляции между присутствием одного или нескольких однонуклеотидных полиморфизмов и присутствия конкретного фенотипического признака - позволяет отслеживать в популяции растений-потомков те растения, которые обладают генетическим потенциалом в проявлении конкретного фенотипического признака. Таким образом, путем обеспечения неограничивающего перечня маркеров объекты, раскрытые в настоящем изобретении, предусматривают эффективное применение раскрытых в настоящем изобретении аллелей и/или гаплотипов, ассоциированных с оптимизацией потребления воды, в селекционных программах. В некоторых вариантах осуществления маркер является специфическим для конкретной генеалогической линии. Таким образом, специфический признак может быть ассоциирован с конкретным маркером.
Раскрытые в данном документе маркеры могут не только указывать на местоположение аллеля, ассоциированного с оптимизацией потребления воды, они также коррелируют с присутствием специфического фенотипического признака у растения. Следует отметить, что однонуклеотидные полиморфизмы, которые указывают на то, где аллель, ассоциированный с оптимизацией потребления воды, присутствует в геноме, являются неограничивающими. Обычно на местоположение аллеля, ассоциированного с оптимизацией потребления воды, указывают с помощью набора однонуклеотидных полиморфизмов, которые проявляют статистическую корреляцию с фенотипическим признаком. Когда маркер выявляют за пределами однонуклеотидного полиморфизма (т.е. он имеет LOD-балл ниже определенного порога, указывающего на то, что маркер является настолько удаленным, что рекомбинация в участке между маркером и аллелем, ассоциированным с оптимизацией потребления воды, происходит настолько часто, что присутствие маркера статистически значимо не коррелирует с присутствием данного фенотипа), границы аллеля, ассоциированного с оптимизацией потребления воды, могут считаться установленными. Таким образом, также можно указать местоположение аллеля, ассоциированного с оптимизацией потребления воды, с помощью других маркеров, расположенных в пределах такого указанного участка. Дополнительно следует отметить, что однонуклеотидный полиморфизм можно также применять для указания присутствия аллеля (и, таким образом, фенотипа), ассоциированного с оптимизацией потребления воды, в отдельном растении, что в некоторых вариантах осуществления означает, что его можно применять в процедурах отбора с помощью маркеров (MAS).
В принципе, число потенциально применимых маркеров может быть очень велико. Любой маркер, который сцеплен с аллелем, ассоциированным с оптимизацией потребления воды (например, находящийся в пределах физических границ участка генома, перекрываемых маркерами, характеризующихся установленными LOD-баллами, которые выше определенного порога, что тем самым указывает на отсутствие рекомбинации или незначительную рекомбинацию между маркером и аллелем, ассоциированным с оптимизацией потребления воды, при скрещиваниях, а также любой маркер, находящийся в неравновесном сцеплении с аллелем, ассоциированным с оптимизацией потребления воды, а также маркеры, которые представляют собой настоящие причинные мутации в пределах аллеля, ассоциированного с оптимизацией потребления воды), можно применять в способах и композициях, раскрытых в настоящем изобретении, и эти маркеры находятся в пределах объема объектов, раскрытых в настоящем изобретении. Это означает, что маркеры, идентифицированные в данной заявке как ассоциированные с аллелем, ассоциированным с оптимизацией потребления воды (например, маркеры, которые присутствуют в SEQ ID NO: 1-8, 17-65 или содержат любую из них, а также и аллели, идентифицированные в таблицах 1-7), представляют собой неограничивающие примеры маркеров, подходящих для применения в способах и композициях, раскрытых в настоящем изобретении. Более того, если аллель, ассоциированный с оптимизацией потребления воды, или его обеспечивающая специфический признак часть интрогрессируются в другое генетическое окружение (т.е. в геном другого растения маиса или другого вида растений), то некоторые маркеры могут больше не обнаруживаться у потомства, хотя признак у них присутствует, что свидетельствует о том, что такие маркеры находятся за пределами участка генома, который представляет собой обеспечивающую специфический признак часть аллеля, ассоциированного с оптимизацией потребления воды, только в исходной родительской линии, и что новое генетическое окружение характеризуется другой организацией генома. Такие маркеры, отсутствие которых указывает на успешное введение генетического элемента в потомство, называются "транс-маркерами", и они, в равной степени, могут быть пригодны для объектов, раскрытых в настоящем изобретении.
После идентификации аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды, влияние аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды (например, признака), например, может быть подтверждено путем оценки признака в потомстве, сегрегирующем по исследуемым аллелям и/или гаплотипам, ассоциированным с оптимизацией потребления воды. Для удобства оценку признака можно проводить с применением фенотипической оценки, как известно из уровня техники для признаков оптимизации потребления воды. Например, для оценки признаков гибридного и/или инбредного маиса можно проводить (полевые) испытания в естественных условиях и/или условиях полива.
Маркеры, обеспечиваемые объектами, раскрытыми в настоящем изобретении, можно применять для выявления присутствия одного или нескольких аллелей и/или гаплотипов признака оптимизации потребления воды в локусах объектов, раскрытых в настоящем изобретении, в растении маиса с предполагаемым интрогрессированным признаком оптимизации потребления воды, и, следовательно, их можно применять в способах, предусматривающих селекцию с помощью маркеров и отбор таких растений маиса, несущих признак оптимизации потребления воды. В некоторых вариантах осуществления выявление присутствия аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды, в объектах, раскрытых в настоящем изобретении, проводят по меньшей мере с помощью одного из маркеров для аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды, определяемых в данном документе. Таким образом, объекты, раскрытые в настоящем изобретении, относятся к другому аспекту способа выявления присутствия аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды, обеспечивающих по меньшей мере один из признаков оптимизации потребления воды, раскрытых в настоящем изобретении, предусматривающего выявление присутствия последовательности нуклеиновой кислоты аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды, в растении маиса, несущем признак, при этом присутствие можно выявлять путем применения раскрытых маркеров.
В некоторых вариантах осуществления выявление предусматривает определение нуклеотидной последовательности нуклеиновой кислоты Zea mays, ассоциированной с признаком, аллелем и/или гаплотипом, ассоциированными с оптимизацией потребления воды. Нуклеотидную последовательность аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды, из объектов, раскрытых в настоящем изобретении, к примеру, можно получить путем определения нуклеотидной последовательности одного или нескольких маркеров, ассоциированных с аллелем и/или гаплотипом, ассоциированными с оптимизацией потребления воды, и конструированием внутренних праймеров для маркерных последовательностей, которые затем можно применять для дальнейшего определения последовательности аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды, за пределами маркерных последовательностей.
Например, нуклеотидная последовательность маркеров SNP, раскрытых в данном документе, может быть получена путем выделения маркеров из электрофоретического геля, используемого для определения присутствия маркеров в геноме заданного растения, и определения нуклеотидной последовательности маркеров, например, с помощью способов секвенирования с обрывом дидезокси-цепи, которые хорошо известны из уровня техники. В некоторых вариантах осуществления таких способов выявления присутствия аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды, в растении маиса, несущем признак, данный способ также может предусматривать обеспечение олигонуклеотида или полинуклеотида, способных к гибридизации в условиях жесткой гибридизации с последовательностью нуклеиновой кислоты маркера, сцепленного с аллелем и/или гаплотипом, ассоциированными с оптимизацией потребления воды, в некоторых вариантах осуществления выбранного из маркеров, раскрытых в данном документе, приведение олигонуклеотида или полинуклеотида в контакт с расщепленной геномной нуклеиновой кислотой растения маиса, несущего признак, и определение присутствия специфической гибридизации олигонуклеотида или полинуклеотида с расщепленной геномной нуклеиновой кислотой. В некоторых вариантах осуществления способ проводится с образцом нуклеиновой кислоты, полученным из растения маиса, несущего признак, хотя также можно использовать способы in situ гибридизации. В качестве альтернативы, после того как была определена нуклеотидная последовательность аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды, средний специалист в данной области может сконструировать специфические гибридизационные зонды или олигонуклеотиды, способные к гибридизации в жестких условиях гибридизации с последовательностью нуклеиновой кислоты аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды, и такие гибридизационные зонды можно применять в способах выявления присутствия раскрытых в данном документе аллеля и/или гаплотипа, ассоциированных с оптимизацией потребления воды, в растении маиса, несущем признак.
Конкретные нуклеотиды, которые присутствуют в конкретных местоположениях в маркерах и нуклеиновых кислотах, раскрытых в данном документе, можно определить с применением методик молекулярной биологии, включая без ограничения амплификацию геномной ДНК из растений и последующее секвенирование. Кроме того, можно сконструировать олигонуклеотидные праймеры, которые, как ожидается, будут специфически гибридизироваться с конкретными последовательностями, которые содержат полиморфизмы, раскрытые в данном документе. Например, можно сконструировать олигонуклеотиды, которые позволят различать аллель "А" и аллель "G" в нуклеотида, которое соответствует положению 401 из SEQ ID NO: 17, с применением олигонуклеотидов содержащих, состоящих фактически из или состоящих из SEQ ID NO: 27 и 28. Соответствующее отличие между SEQ ID NO: 27 и 28 состоит в том, что первая имеет нуклеотид G в положении 15, а последняя имеет А нуклеотид в положении 16. Таким образом, можно сконструировать условия гибридизации для SEQ ID NO: 27, которые будут позволять SEQ ID NO: 27 специфически гибридизироваться с аллелем "G", в случае его присутствия, но не с аллелем "А", в случае его присутствия. Таким образом, гибридизацию с применением двух данных праймеров, которые отличаются только одним нуклеотидом, можно использовать для анализа в отношении присутствия одного или другого аллеля в положении нуклеотида, которое соответствует 401 из SEQ ID NO: 17.
В некоторых вариантах осуществления маркер может содержать, состоять фактически из или состоять из обратной комплементарной последовательности любого из упомянутых выше маркеров. В некоторых вариантах осуществления один или несколько аллелей, которые составляют маркерный гаплотип, присутствуют, как описано выше, тогда как один или несколько других аллелей, которые составляют маркерный гаплотип, присутствуют в виде обратной комплементарной последовательности описанного выше аллеля(-ей). В некоторых вариантах осуществления каждый из аллелей, которые составляют маркерный гаплотип, присутствуют в виде обратной комплементарной последовательности описанного выше аллеля(-ей).
В некоторых вариантах осуществления маркер может содержать, состоять фактически из или состоять из информативного фрагмента любого из упомянутых выше маркеров, обратной комплементарной последовательности любого из упомянутых выше маркеров или информативного фрагмента обратной комплементарной последовательности любого из упомянутых выше маркеров. В некоторых вариантах осуществления один или несколько аллелей/последовательностей, которые составляют маркерный гаплотип, присутствуют как описано выше, тогда как один или несколько других аллелей/последовательностей, которые составляют маркерный гаплотип, присутствуют в виде обратной комплементарной последовательности описанных выше аллелей/последовательностей. В некоторых вариантах осуществления один или несколько аллелей/последовательностей, которые составляют маркерный гаплотип, присутствуют как описано выше, тогда как один или несколько других аллелей/последовательностей, которые составляют маркерный гаплотип, присутствуют в виде информативного фрагмента описанных выше аллелей/последовательностей. В некоторых вариантах осуществления один или несколько аллелей/последовательностей, которые составляют маркерный гаплотип, присутствуют как описано выше, тогда как один или несколько других аллелей/последовательностей, которые составляют маркерный гаплотип, присутствуют в виде информативного фрагмента обратной комплементарной последовательности описанных выше аллелей/последовательностей. В некоторых вариантах осуществления каждый из аллелей/последовательностей, которые составляют маркерный гаплотип, присутствует в виде информативного фрагмента описанных выше аллелей/последовательностей, обратной комплементарной последовательности описанных выше аллелей/последовательностей или информативного фрагмента обратной комплементарной последовательности описанных выше аллелей/последовательностей.
В некоторых вариантах осуществления маркер может содержать, состоять фактически из или состоять из любого маркера, сцепленного с упомянутыми выше маркерами. То есть любой аллель и/или гаплотип, который находится в неравновесном сцеплении с любым из упомянутых выше маркеров, можно также применять для идентификации, отбора и/или получения растения маиса с повышенной засухоустойчивостью. Сцепленные маркеры можно определять, например, с применением ресурсов, доступных на веб-сайте MaizeGDB.
Также предусмотрены выделенные и очищенные маркеры, ассоциированные с повышенной засухоустойчивостью. Такие маркеры могут содержать, состоять фактически из или состоять из нуклеотидной последовательности, изложенной под любой из SEQ ID NO: 1-8 и 17-65, аллелей, описанных в таблицах 1-7 и их обратных комплементарных последовательностей или их информативного фрагмента. В некоторых вариантах осуществления маркер содержит выявляемый фрагмент. В некоторых вариантах осуществления маркер позволяет выявлять один или несколько маркерных аллелей, идентифицированных в данном документе.
Также предусмотрены композиции, содержащие пару праймеров, которые способны амплифицировать образец нуклеиновой кислоты, выделенный из растения или идиоплазмы маиса, с получением маркера, ассоциированного с повышенной засухоустойчивостью. В некоторых вариантах осуществления маркер содержит нуклеотидную последовательность, изложенную в данном документе, ее обратную комплементарную последовательность или ее информативный фрагмент. В некоторых вариантах осуществления маркер содержит нуклеотидную последовательность, которая по меньшей мере на приблизительно 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% 97%, 99% или 100% идентична нуклеотидной последовательности, изложенной в данном документе, ее обратной комплементарной последовательности или ее информативному фрагменту. В некоторых вариантах осуществления пара праймеров представляет собой одну из пар праймеров для амплификации, идентифицированных в таблице 8 выше. Средний специалист в данной области будет понимать, как выбрать альтернативные пары праймеров в соответствии со способами, хорошо известными из уровня техники.
Идентификация растений с разными представляющими интерес аллелями и/или гаплотипами может обеспечить исходные материалы для комбинирования аллелей и/или гаплотипов в растениях-потомках посредством стратегий селекции, разработанных для "пакетирования" аллелей и/или гаплотипов. Используемый в данном документе термин "пакетирование" и его грамматические варианты относятся к преднамеренному накоплению с помощью селекции (включая без ограничения скрещивание двух растений, самоопыление одного растения и/или создание двойного гаплоида из одного растения) благоприятных гаплотипов оптимизации потребления воды в растениях, вследствие чего геном растения имеет по меньшей мере один дополнительный благоприятный гаплотип оптимизации потребления воды в отличие от его прямого предка(-ов). В некоторых вариантах осуществления пакетирование включает передачу одного или несколько признаков, аллелей и/или гаплотипов оптимизации потребления воды растению-потомку маиса, вследствие чего растение-потомок маиса включает большее число признаков, аллелей и/или гаплотипов оптимизации потребления воды, чем любой из родителей, от которых оно было получено. В качестве примера, а не ограничения, если родитель 1 имеет гаплотипы А, В и С, а родитель 2 имеет гаплотипы D, Е и F, то "пакетирование" относится к получению растения, которое имеет любой из А, В и С в любой комбинации с D, Е и F. В частности, в некоторых вариантах осуществления "пакетирование" относится к получению растения, которое имеет А, В и С, а также один или несколько из D, Е и F, или к получению растения, которое имеет D, Е и F, а также один или несколько из А, В и С. В некоторых вариантах осуществления "пакетирование" относится к получению в результате двуродительского скрещивания растения, в котором содержатся все гаплотипы, ассоциированные с оптимизацией потребления воды, которыми обладают каждый из родителей.
III. Способы интрогрессии представляющих интерес аллелей и идентификации растений, содержащих их
III.А. Принцип отбора с помощью маркеров
Маркеры можно применять в целом ряде применений, связанных с селекцией растений. См., например, Staub et al., Hortscience 31: 729 (1996); Tanksley, Plant Molecular Biology Reporter 1: 3 (1983). Одной из основных представляющих интерес областей является повышение эффективности возвратного скрещивания и интрогрессии генов с применением отбора с помощью маркера (MAS). В целом, в MAS используются генетические маркеры, которые были идентифицированные как характеризующиеся значительной вероятностью косегрегации с требуемым признаком. Предполагается, что такие маркеры находятся в/вблизи гена(-ов), который обуславливает требуемый фенотип, и их присутствие указывает на то, что растение будет обладать требуемым признаком. Ожидается, что растения, которые обладают маркером, передадут требуемый фенотип своему потомству.
Маркер, который демонстрирует сцепление с локусом, воздействующим на требуемый фенотипический признак, обеспечивает применимый инструмент для отбора по данному признаку в популяции растений. Это особенно актуально, если фенотип сложно анализировать или это происходит на поздней стадии развития растения. Так как анализы на основе маркеров ДНК являются менее трудоемкими и занимают меньше физического пространства, чем фенотипирование в полевых условиях, можно проанализировать намного более крупные популяции, что увеличивает шансы обнаружения рекомбинанта с целевым сегментом из линии-донора, перенесенной в линию-реципиента. Чем более тесным является сцепление, тем больше пригоден маркер, поскольку менее вероятна рекомбинация между маркером и геном, вызывающим развитие признака или оказывающим на него влияние. Наличие фланкирующих маркеров снижает шансы того, что будет происходить ложноположительный отбор. Идеальной ситуацией является наличие маркера в самом гене, чтобы не могла происходить рекомбинация между маркером и геном. Такой маркер называют "совершенным маркером".
При интрогрессии гена с помощью MAS вводится не только ген, но также и фланкирующие участки. Gepts, Crop Sci 42:1780 (2002). Их называют "сцепленным грузом". В случае, если растение-донор характеризуется далеким родством с растением-реципиентом, эти фланкирующие участки несут дополнительные гены, которые могут кодировать признаки, нежелательные с точки зрения сельского хозяйства. Этот "сцепленный груз" также может приводить к снижению урожайности или другим отрицательным с точки зрения сельского хозяйства характеристикам даже после нескольких циклов возвратного скрещивания с элитной линией маиса. Иногда его называют также "грузом для урожая". Размер фланкирующего участка можно уменьшать путем дополнительного возвратного скрещивания, хотя это не всегда приносит успех, поскольку селекционеры не контролируют размера участка или точечные разрывы при рекомбинации. Young et al., Genetics 120:579 (1998). В случае классической селекции рекомбинации, которые вносят вклад в уменьшение размера донорного сегмента, обычно происходят лишь случайным образом. Tanksley et al., Biotechnology 7: 257 (1989). Даже после 20 возвратных скрещиваний можно ожидать обнаружение того, что значительная часть донорной хромосомы все еще сцеплена с геном, по которому проводят отбор. Однако с помощью маркеров можно отбирать тех редких особей, у которых произошла рекомбинация вблизи представляющего интерес гена. Среди 150 растений, полученных в результате возвратного скрещивания, существует 95% вероятность того, что по меньшей мере у одного растения произошел кроссинговер в пределах 1 сМ от гена, на основании расстояния по генетической карте одиночного мейоза. Маркеры обеспечивают возможность однозначной идентификации таких особей. При одном дополнительном возвратном скрещивании у 300 растений будет 95% вероятность кроссинговера в пределах 1 сМ расстояния по генетической карте одиночного мейоза с другой стороны гена, что приведет к образованию сегмента вокруг целевого гена размером менее 2 сМ, на основании расстояния по генетической карте одиночного мейоза. С помощью маркеров этого можно достичь за два поколения, в то время как без маркеров потребуется в среднем 100 поколений. См. Tanksley et al., выше. Если известно точное местоположение гена, то для отбора в отношении рекомбинаций в популяциях различного размера можно использовать фланкирующие маркеры, окружающие данный ген. Например, в популяциях меньшего размера рекомбинации можно ожидать дальше от гена, поэтому для выявления рекомбинации будут требоваться более отдаленные фланкирующие маркеры.
Доступность интегрированных карт сцепления для генома маиса, которые содержат все возрастающее число общедоступных маркеров маиса, облегчает генетическое картирование и MAS у маиса. См., например, карты IBM2 Neighbors, которые доступны онлайн на веб-сайте MaizeGDB.
Из всех типов молекулярных маркеров SNP являются наиболее многочисленными и обладают потенциалом обеспечения самого высокого разрешения генетической карты. Bhattramakki et al., Plant Molec. Biol. 48:539 (2002). SNP можно анализировать в так называемом режиме "сверхпроизводительности", поскольку для них не требуются большие количества нуклеиновой кислоты, а автоматизация анализа является весьма простой. SNP также обладают преимуществом в том, что являются относительно недорогими системами. Вместе эти три фактора делают SNP весьма привлекательными для применения в MAS. Для генотипирования SNP доступны несколько способов, включая без ограничений гибридизацию, удлинение праймера, лигирование олигонуклеотида, расщепление под действием нуклеаз, минисеквенирование и кодируемые сферы. Обзор таких способов был приведен в различных публикациях: Gut, Hum. Mutat. 17:475 (2001); Shi, Clin. Chem. 47:164 (2001); Kwok, Pharmacogenomics 1:95 (2000); Bhattramakki and Rafalski, Discovery and application of single nucleotide polymorphism markers in plants, в PLANT GENOTYPING: THE DNA FINGERPRINTING OF PLANTS, CABI Publishing, Wallingford (2001). Эти и другие способы детального исследования SNP используются в широком спектре коммерчески доступных методик, включая Masscod™ (Qiagen, Джермантаун, Мэриленд), Invader® (Hologic, Мэдисон, Висконсин), SnapShot® (Applied Biosystems, Фостер-Сити, Калифорния), Taqman® (Applied Biosystems, Фостер-Сити, Калифорния) и Beadarrays™ (Illumina, Сан-Диего, Калифорния).
Для описании гаплотипа в случае любого конкретного генотипа целый ряд SNP можно применять совместно в пределах последовательности или на протяжении сцепленных последовательностей. Ching et al., ВМС Genet. 3:19 (2002); Gupta et al., (2001), Rafalski, Plant Sci. 162:329 (2002b). Гаплотипы могут быть более информативными, чем одиночные SNP, и они могут быть более наглядными, чем любой конкретный генотип. Например, одиночный SNP может представлять собой аллель "Т" в специфической засухоустойчивой линии или сорте, однако аллель "Т" также может встречаться в популяции для селекции маиса, используемой в качестве рекуррентных родителей. В таком случае комбинация аллелей в сцепленных SNP может быть более информативной. После того как уникальный гаплотип связали с хромосомным участком донора, такой гаплотип можно применять в такой популяции или любой ее субпопуляции для определения того, имеет ли особь конкретный ген. Применение автоматизированных высокопроизводительных платформ для обнаружения маркеров, известных средних специалистам в данной области, делает этот способ высокоэкономичным и эффективным.
Маркеры согласно объектам, раскрытым в настоящем изобретении, можно применять в протоколах отбора с помощью маркеров для идентификации и/или отбора потомства с повышенной засухоустойчивостью. Такие способы могут предусматривать, включать фактически или включать скрещивание первого растения или идиоплазмы маиса со вторым растением или идиоплазмой маиса, где первое растение или идиоплазма маиса содержат маркер, ассоциированный с повышенной засухоустойчивость, и отбор растения-потомка, которое имеет данный маркер. Любое из первого и второго растений маиса или они оба могут представлять собой не встречающийся в природе сорт маиса.
III.В. Способы интрогрессии представляющих интерес аллелей и/или гаплотипов
Таким образом, в некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, предусмотрены способы интрогрессии аллеля, ассоциированного с повышенной засухоустойчивостью, в генетическое окружение, не имеющее указанного аллеля. В некоторых вариантах осуществления способ предусматривает скрещивание донора, содержащего указанный аллель, с рекуррентным родителем, у которого отсутствует указанный аллель; и повторное возвратное скрещивание потомков, содержащего указанный аллель, с рекуррентным родителем, где указанных потомков идентифицируют путем выявления в их геномах присутствия маркера в пределах хромосомного интервала из группы, состоящей из:
(a) хромосомного интервала на хромосоме 1, определяемого от положения пары оснований (п. о.) 272937470 до положения пары оснований (п. о.) 272938270 включительно (PZE01271951242);
(b) хромосомного интервала на хромосоме 2, определяемого от положения пары оснований (п. о.) 12023306 до положения пары оснований (п. о.) 12024104 включительно (PZE0211924330);
(c) хромосомного интервала на хромосоме 3, определяемого от положения пары оснований (п. о.) 225037202 до положения пары оснований (п. о.) 225038002 включительно (PZE03223368820);
(d) хромосомного интервала на хромосоме 3, определяемого от положения пары оснований (п. о.) 225340531 до положения пары оснований (п. о.) 225341331 включительно (PZE03223703236);
(е) хромосомного интервала на хромосоме 5, определяемого от положения пары оснований (п. о.) 159120801 до положения пары оснований (п. о.) 159121601 включительно (PZE05158466685);
(f) хромосомного интервала на хромосоме 9, определяемого от положения пары оснований (п. о.) 12104536 до положения пары оснований (п. о.) 12105336 включительно (PZE0911973339);
(g) хромосомного интервала на хромосоме 9, определяемого от положения пары оснований (п. о.) 225343590 до положения пары оснований (п. о.) 225340433 включительно (S_l8791654);
(h) хромосомного интервала на хромосоме 10, определяемого от положения пары оснований (п. о.) 14764415 до положения пары оснований (п. о.) 14765098 (S_20808011); с получением тем самым засухоустойчивого растения или идиоплазмы маиса, содержащих указанный аллель, ассоциированный с повышенной засухоустойчивостью, в генетическом окружении рекуррентного родителя, с интрогрессией тем самым аллеля, ассоциированного с повышенной засухоустойчивостью, в генетическое окружение, не имеющее указанного аллеля. В некоторых вариантах осуществления геном указанного засухоустойчивого растения или идиоплазмы маиса, содержащий указанный аллель, ассоциированный с повышенной засухоустойчивостью, является по меньшей мере на приблизительно 95% идентичным геному рекуррентного родителя. В некоторых вариантах осуществления любой из донора или рекуррентного родителя или они оба представляют собой не встречающийся в природе сорт маиса.
Таким образом, в некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, предусмотрен способ получения растения с повышенной урожайностью, предусматривающий стадии:
a. отбора из разнородной популяции растений с применением маркера, выбранного из группы, состоящей из маркеров SM2973, SM2980, SM2982, SM2984, SM2987, SM2991, SM2995, SM2996;
b. размножения/самоопыления растения.
В дополнительных вариантах осуществления способа согласно объектам, раскрытым в настоящем изобретении, предусмотрен способ получения растения с повышенной урожайностью, предусматривающий стадии:
а. отбора из разнородной популяции растений с применением маркера, выбранного из группы, состоящей из маркеров SM2973, SM2980, SM2982, SM2984, SM2987, SM2991, SM2995, SM2996; где
маркер SM2973 имеет "G" в нуклеотиде 401;
маркер SM2980 имеет "С" в нуклеотиде 401;
маркер SM2982 имеет "А" в нуклеотиде 401;
маркер SM2984 имеет "G" в нуклеотиде 401;
маркер SM2987 имеет "G" в нуклеотиде 401;
маркер SM2991 имеет "G" в нуклеотиде 401;
маркер SM2995 имеет "А" в нуклеотиде 401 и
маркер SM2996 имеет "А" в нуклеотиде 401.
III.D. Способы пакетирования представляющих интерес аллелей и/или гаплотипов
В некоторых вариантах осуществления объекты, раскрытые в настоящем изобретении, относятся к "пакетированию" гаплотипов, ассоциированных с оптимизацией потребления воды с целью получения растений (и их частей), которые имеют несколько благоприятных локусов оптимизации потребления воды. В качестве примера, а не ограничения, в некоторых вариантах осуществления объекты, раскрытые в настоящем изобретении, относятся к идентификации и определению характеристик локусов Zea mays, каждый из которых ассоциирован с одним или несколькими признаками оптимизации потребления воды. Данные локусы соответствуют SEQ ID NO: 1-8 и 17-65, а также имеют гаплотипы А-М, определенный в данном документе.
Для каждого из этих локусов были идентифицированы благоприятные аллели, которые ассоциированы с признаками оптимизации потребления воды. Эти благоприятные аллели кратко изложены в данном документе, например, в таблицах 1-7, или любые маркеры, тесно сцепленные с генами, перечислены в таблице 9. Объекты, раскрытые в настоящем изобретении, предусматривают иллюстративные аллели (например, показанные в таблицах 1-7 или таблице 11), которые ассоциированы с повышением и понижением разных признаков оптимизации потребления воды, определенных в данном документе.
III.Е. Способы идентификации растений, содержащих представляющие интерес аллели и/или гаплотипы
Способы идентификации засухоустойчивого растения или идиоплазмы маиса могут предусматривать выявление присутствия маркера, ассоциированного с повышенной засухоустойчивостью. Маркер можно выявлять в любом образце, полученном из растения или идиоплазмы, включая без ограничения целое растение или идиоплазму, часть указанного растения или идиоплазмы (например, клетку из указанного растения или идиоплазмы) или нуклеотидную последовательность из указанного растения или идиоплазмы. Растение маиса может представлять собой не встречающийся в природе сорт маиса. В некоторых вариантах осуществления геном растения или идиоплазма маиса по меньшей мере на приблизительно 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 97%, 99% или 100% идентичны геному элитного сорта маиса.
Способы интрогрессии аллеля, ассоциированного с повышенной засухоустойчивый, в растение или идиоплазму маиса могут предусматривать скрещивание первого растения или идиоплазмы маиса, содержащих указанный аллель (донор), со вторым растением или идиоплазмой маиса, в которых отсутствует указанный аллель (рекуррентный родитель), и повторное возвратное скрещивание потомства, содержащего указанный аллель, с рекуррентным родителем. Потомков, содержащих указанный аллель, можно идентифицировать путем выявления в их геномах присутствия маркера, ассоциированного с повышенной засухоустойчивостью. Любой из донора или рекуррентного родителя или они оба могут представлять собой не встречающийся в природе сорт маиса.
IV. Получение растений маиса, несущих улучшенный признак, с помощью трансгенных способов
В некоторых вариантах осуществления объекты, раскрытые в настоящем изобретении, связаны с применением полиморфизмов (включая без ограничения SNP) или обеспечивающих признак частей для получения несущего признак растения маиса путем введения последовательности нуклеиновой кислоты, содержащей ассоциированный с признаком аллель и/или гаплотип полиморфизма, в растение-реципиент.
Растение-донор с последовательностью нуклеиновой кислоты, которая содержит аллель и/или гаплотип признака оптимизации потребления воды, можно переносить в растение-реципиента, в котором отсутствует данный аллель и/или гаплотип. Последовательность нуклеиновой кислоты можно переносить путем скрещивания растения-донора, несущего признак оптимизации потребления воды, с растением-реципиентом, которое не несет признак (например, путем интрогрессии), с помощью трансформации, с помощью трансформации или слияния протопластов, с помощью методики двойного гаплоида, с помощью эмбрионального спасения или с помощью любой другой системы переноса нуклеиновой кислоты. Далее, при необходимости, можно провести отбор растений-потомков, содержащих один или несколько аллелей и/или гаплотипов оптимизации потребления воды, раскрытых в настоящем изобретении. Последовательность нуклеиновой кислоты, содержащую аллель и/или гаплотип признака оптимизации потребления воды, можно выделить из растения-донора с применением способов, известных из уровня техники, и выделенными последовательностями нуклеиновой кислоты можно трансформировать растение-реципиент с помощью трансгенных способов. Это может происходить с помощью вектора, в гамете, или с применением другого подходящего элемента для переноса, как, например, баллистической частицы, с нанесенной на нее последовательностью нуклеиновой кислоты.
Трансформация растений обычно включает конструирование вектора экспрессии, который будет функционировать в растительных клетках, и он содержит последовательность нуклеиновой кислоты, которая содержит аллель и/или гаплотип, ассоциированные с признаком оптимизации потребления воды, причем вектор может содержать ген, придающий признак оптимизации потребления воды. Данный ген обычно контролируется одним или несколькими регуляторными элементами, такими как промотор, или функционально связан с ними. Вектор экспрессии может содержать один или несколько таких функционально связанных комбинаций ген/регуляторный элемент при условии, что по меньшей мере один из генов, содержащихся в комбинациях, кодирует признак оптимизации потребления воды. Вектор(-ы) могут быть в виде плазмиды, и их можно использовать отдельно или в комбинации с другими плазмидами для получения трансгенных растений, которые представляют собой растения с улучшенной оптимизацией потребления воды, с применением известных из уровня техники способов трансформации, таких как система для трансформации на основе Agrobacterium. В некоторых вариантах осуществления настоящего изобретения гены, содержащиеся в хромосомных интервалах по данному документу, можно трансгенно экспрессировать в растениях с получением растений с повышенной засухоустойчивостью; кроме того, без ограничения теорией генные модели, показанные в таблице 9, можно трансгенно экспрессировать в растениях с получением растений с повышенной засухоустойчивостью.
Трансформированные клетки зачастую содержат селектируемый маркер для обеспечения возможности идентификации трансформации. Селектируемый маркер, как правило, адаптирован таким образом, чтобы восстанавливаться с помощью отрицательного отбора (путем подавления роста клеток, которые не содержат селектируемый маркерный ген), или с помощью положительного отбора (путем скрининга в отношении продукта, кодируемого селектируемым маркерным геном). Многие селектируемые маркерные гены, широко используемые для трансформации растений, хорошо известны из уровня техники, и они включают, например, гены, которые кодируют ферменты, которые метаболически обезвреживают селективное химическое средство, которое может представлять собой антибиотик или гербицид, или гены, которые кодируют измененную мишень, которая не чувствительна к ингибитору. Из уровня техники известно несколько способов положительного отбора, таких как отбор с помощью маннозы. В качестве альтернативы может применяться трансформация без маркера с получением растений без упомянутых выше маркерных генов, методики которой известны из уровня техники.
Гены оптимизации потребления воды
Множественные положительные ассоциации в анализе SM2987 с повышенной урожайностью в условиях засухи идентифицируют ген GRMZM2G027059 в качестве гена оптимизации потребления воды. GRMZM2G027059 кодирует 4-гидрокси-3-метилбут-2-енилдифосфатредуктазу, которая представляет собой последний фермент в биосинтезе изопентилдифосфата (IPP) и диметилаллилдифосфата (DMAPP) (Arturo Guevara-Garci'a,The Plant Cell, Vol. 17, 628-643), February 2005. У высших растений для синтеза основных изопреноидных звеньев используются два пути. Мевалоновый (MVA) путь происходит в цитоплазме, где образуются сесквитерпены (С15) и тритерпены (С30), такие как фитостеролы, долихолы и остатки фарнезила, для пренилирования белков, а метил-D-эритритол-4-фосфатный (МЕР) путь осуществляется в пластидах, и в ходе него образуются IPP и DMAPP для синтеза изопреноидов, таких как изопрен, каротиноиды, пластохиноны, конъюгаты фитола (такие как хлорофиллы и токоферолы) и гормоны (гиббереллины и абсцизиновая кислота). Имеются указания на то, что между этими двумя путями существует перекрестное взаимодействие (Hsieh and Goodman. Plant Physiology, June 2005). Поскольку GRMZM2G027059 кодирует 4-гидрокси-3-метилбут-2-енилдифосфатредуктазу, которая является важнейшим ферментом для биосинтеза фотопигментов, таких как хлорофиллы и каротиноиды, а также гормонов, таких как гиббереллины и абсцизиновая кислота, растения, экспрессирующие этот ген, могут быть более устойчивыми к абиотическому стрессу.
Множественные положительные ассоциации в анализе SM2991 с повышенной урожайностью в условиях засухи идентифицируют ген GRMZM2G156365 в качестве гена оптимизации потребления воды. GRMZM2G156365 относится к семейству пектинацетилэстераз (РАЕ). Пектинацетилэстеразы катализируют деацетилирование пектина, основного компонента первичных клеточных стенок. Данные, полученные с помощью проприетарного экспрессионного чипа, показывают, что GRMZM2G156365 характеризуется высоким уровнем экспрессии в пыльце и пыльниках, и GRMZM2G156365 характеризовался более высоким уровнем экспрессии в засухоустойчивом гибриде маиса, чем в чувствительном к засухе гибриде маиса. Растения табака, сверхэкспрессирующие РАЕ тополя, PtPAE, проявляют сильную мужскую стерильность, которая затрудняет прорастание пыльцы и удлинение пыльцевой трубки, поэтому растения производят небольшое количество зрелых семян или совсем не производят их (Gou, J.Y., L.M. Miller, et al. (2012). "Acetylesterase-mediated deacetylation of pectin impairs cell elongation, pollen germination, and plant reproduction." Plant Cell 24(1): 50-65). Потеря урожая, вызванная стерильностью пыльцы, является одной из главных проблем, вызываемых засухой. Прорастание пыльцы и удлинение пыльцевой трубки требует точного состояния ацетилирования пектина в клеточной стенке. GRMZM2G156365 может функционировать в качестве регулятора структуры за счет модуляции точного состояния ацетилирования пектина, чтобы воздействовать на ремоделирование и физико-химические свойства клеточной стенки, с воздействием тем самым на способность к растяжению пыльцевой клетки. У растений, в которых снижена экспрессии гена GRMZM2G156365, может повышаться прорастание пыльцы в условиях абиотических стрессов, таких как засуха.
Множественные положительные ассоциации в анализе SM2995 с повышенной урожайностью в условиях засухи идентифицируют ген GRMZM2G134234 в качестве гена оптимизации потребления воды. GRMZM2G134234 содержат домен IPR012866, белка DUF1644 с неизвестной функцией. Данное семейство состоит из последовательностей, найденных в ряде гипотетических белков растений с неизвестной функцией. Представляющий интерес участок содержит девять высококонсервативных остатков цистеина, и его длина составляет примерно 160 аминокислот, по-видимому, он представляет собой цинк-связывающий домен. Ген, кодирующий DUF1644 Arabidopsis, AT3G25910, реагирует на обработку GA и ABA (Guo, С. et al., J Integr Plant Biol (2015)). Имеется 9 представителей семейства DUF1644 риса, которые могут быть вовлечены в реакцию на стресс. SIDP364 локализован в ядре, и он индуцировался под действием ABA, высокой концентрации солей, засухи, тепла, холода и Н2О2. Сверхэкспрессия в рисе повышает чувствительность к ABA и устойчивость к высокой засоленности (вследствие накопления пролина и повышения экспрессии генов, обеспечивающих реакцию на стресс). SIDP361 обладает функцией, аналогичной SIDP364, в случае стресс, вызванного засолением, за счет регулирования АВА-зависимого или независимого сигнального пути. Однако они характеризуются разной реакций на разные стрессы (REF). Семейство DUF1644-содержащих генов может регулировать реакции на абиотический стресс у риса. Сверхэкспрессия OsSIDP366 в рисе повышала засухоустойчивость и устойчивость к засолению и снижала потерю воды, и при этом растения с РНК-интерференцией были более чувствительными к обработкам с применением засоленности и засухи (Guo, С., С. Luo, et al. (2015). "OsSIDP366, a DUF1644 gene, positively regulates responses to drought and salt stresses in rice." J Integr Plant Biol). DUF1644-содержащие гены могут регулировать реакции на абиотические стрессы. GRMZM2G134234 мог положительно регулировать гены, обеспечивающие реакцию на стресс, для повышения стрессоустойчивости маиса. Растения, сверхэкспрессирующие GRMZM2G134234, могли быть более устойчивыми к абиотическим стрессам, таким как засуха и засоленность.
Множественные положительные ассоциации в анализе SM2996 с повышенной урожайностью в условиях засухи идентифицируют ген GRMZM2G094428 в качестве гена оптимизации потребления воды. GRMZM2G094428 содержит домен хлорамфениколтрансферазы IPR003480. Ацилирование представляет собой обычную и биохимически значимую модификацию вторичных метаболитов растений. Большое семейство ацилтрансфераз под названием BAHD утилизируют сложные СоА-тиоэфиры и катализируют образование разных групп метаболитов растений. Суперсемейство BAHD содержит обширную группу ферментов, характеризующихся незначительным сходством аминокислотных последовательностей, но имеющих два консенсусные мотивы, HXXXD и DFGWG. GRMZM2G094428 филогенетически наиболее подобна BAD-трансферазам, вовлеченным в ферулоилирование/кумароилирование клеточной стенки. Ожидается, что GRMZM2G094428 вовлечен в ферулоилирование/кумароилирование клеточной стенки. Клеточные стенки травянистых растений, таких как пшеница, маис, рис и сахарный тростник, содержат два наиболее заметных соединения, которые представляют собой р-кумаровую кислоту (рСА) и феруловую кислоту (FA). рСА практически полностью этерифицируется до лигнина, a FA этерифицируется до GAX в клеточной стенке (Lu and Ralph, 1999). Было идентифицировано, что суперсемейство ацил-соА-трансфераз BAHD является ответственным за данный процесс (Hugo, et al., 2013). Сверхэкспрессия или нокаут ацил-соА-трансферазы BAHD могли менять состав клеточной стенки. Нокаут ацил-соА-трансферазы BAHD мог снижать содержание FA или р-СА, изменять содержание лигнина (Piston et al., 2010). Сверхэкспрессия OsAT10 в рисе может повышать содержание связанной со сложным эфиром р-СА, ассоциированной с полисахаридами матрикса, при одновременном снижении FA, ассоциированной с полисахаридами матрикса, но при отсутствии выраженных фенотипических изменений в вегетативном развитии, содержании лигнина или составе лигнина (Larua et al., 2013). У линии с РНК-интерференцией рСАТ было показан сниженный уровень рСА, но уровни лигнина не изменились (Jane, et al., 2014) Lignin and abiotic stress (обзорная статья Michael, 2013). Лигнификация тканей культурных растений воздействует на выживаемость растений и может придавать устойчивость к абиотическим стрессам. Для трансгенных растений табака с повышенными уровнями лигнина показана улучшенная засухоустойчивость по сравнению с диким типом. Мутанты маиса с дефицитом лигнина проявляли признаки поражения засухой даже в условиях достаточного количества воды, и при этом в наборе различающихся генотипов уровни лигнина в листьях коррелировали с засухоустойчивостью. Линия трансгенного риса, в корнях которого накапливались повышенные уровни лигнина, при обработке с засоленностью была более устойчивой, чем дикий тип, который не демонстрировал такую реакцию. GRMZM2G094428 мог быть ответственным за р-кумароилирование монолигнолов, которые в конечном счете вовлечены в биосинтез лигнина, а также ответственны за этерификацию FA до GAX в клеточной стенке. Повышенное содержание лигнина может придавать растению устойчивость в условиях абиотических стрессов, включая засуху и засоленность.
Множественные положительные ассоциации в анализе SM2973 с повышенной урожайностью в условиях засухи идентифицируют ген GRMZM2G416751 в качестве гена оптимизации потребления воды. GRMZM2G416751 характеризуется 62% идентичностью и 83% сходством с 450 аминокислотами на С-конце гена AT5G58100.1 Arabidopsis. В линиях мутантов spot1 (SALK_061320, SALK_041228 и SALK_079847) At5g58100 был нарушен за счет вставок Т-ДНК в разных участках. Элементы экзины у мутанта spot1, по-видимому, являются почти полностью разъединенными, что указывает на возможные проблемы с образованием тектума (Dobritsa, А.А., А. Geanconteri, et al. (2011). "A large-scale genetic screen in Arabidopsis to identify genes involved in pollen exine production." Plant Physiol 157(2): 947-970). Потеря урожая, вызванная стерильностью пыльцы, является одной из главных проблем, вызываемых засухой. GRMZM2G416751 мог быть вовлечен в образование экзины пыльцы с повышением стрессоустойчивости маиса. Растения, сверхэкспрессирующие данный ген, могли избегать стерильности пыльцы в условиях стресса, вызванного засухой.
Множественные положительные ассоциации в анализе SM2980 с повышенной урожайностью в условиях засухи идентифицируют ген GRMZM2G467169 в качестве гена оптимизации потребления воды. GRMZM2G467169 имеет предсказанный консервативный домен семейства полиаденилат-связывающих белков человеческого типа. GRMZM2G467169 экспрессируется на высоком уровне в тканях листьев и репродуктивных тканях. AT4G01290 (RIMB3), предполагаемый ортолог в Arabidopsis, положительно регулирует 2СРА (2-цис-пероксиредоксин А) при ретроградной передаче окислительно-восстановительного сигнала от хлоропласта к ядру. Мутант rimb3 рос медленнее и имел листья меньшего размера, а у более крупных растений rimb3 имелся хлороз в условиях длинного дня. В растительных клетках RIMB3 играет роль сенсора при реакции на биотический или абиотический стрессы. Белок AT4G01290 связывается с комплексом 5'-кэпа в Arabidopsis. AT4G01290 взаимодействует с UBQ3 и возможно разрушается под действием 26S протеасомы. В условиях различных биотических и абиотических стрессов сигналы, такие как окислительно-восстановительный дисбаланс в PS1, возникающие в хлоропласте, передаются в ядро для воздействия на паттерн экспрессии генов (ретроградная передача сигнала). GRMZM2G467169 может регулировать ретроградную передачу сигналов для повышения стрессоустойчивости маиса. Растения, в которых сверхэкспрессируется данный ген, могут быть более устойчивыми к абиотическим стрессам, таким как засуха.
Множественные положительные ассоциации в анализе SM2982 с повышенной урожайностью в условиях засухи идентифицируют ген GRMZM5G862107 в качестве гена оптимизации потребления воды. GRMZM5G862107 содержит РНК-связывающий домен, S1, IPR006196, и характеризуется 69% идентичностью с белком AT5G30510 из Arabidopsis. Домен S1 имеет высокую степень сходства с таким доменом белка холодового шока (Bycroft et al., Cell, January 1997). Белки холодового шока (CSP) содержат РНК-связывающие последовательности, обозначаемые как домены холодового шока (CSD), и они хорошо известны своим действием в качестве шаперонов для РНК. Роль CSP у бактерий заключается в адаптации к холодовому стрессу. CSD-содержащие белки растений имеют высокий уровень сходства с CSP бактерий, и было показано, что они имеют общие in vitro и in vivo функции с CSP бактерий (Journal of Experimental Botany, Vol. 62, No. 11, pp. 4003-4011, 2011). CSD-содержащие белки растений, как сообщалось, обычно обеспечивают реакцию на абиотические стрессы. Растения, в которых сверхэкспрессируется данный ген, могут быть более устойчивыми к абиотическим стрессам, таким как засуха.
Множественные положительные ассоциации в анализе SM2984 с повышенной урожайностью в условиях засухи идентифицируют ген GRMZM2G050774 в качестве гена оптимизации потребления воды. GRMZM2G050774 кодирует домен белка RING-палец подтипа Н2 (С3НС4), предположительно Е3-лигазу. Сообщалось, что Е3-лигазы, такие как ATL31/6 у Arabidopsis, функционируют в регуляции метаболизма углерода и азота (Plant Signal Behav. 2011 Oct; 6(10): 1465-1468). GRMZM2G050774 может быть вовлечен в путь передачи сигнала при стрессе, который ответственный за улучшение засухоустойчивости.
Трансформация
Ген хлорамфениколацетилтрансферазы (Callis et al. 1987, Genes Develop.1: 1183-1200). В той же экспериментальной системе интрон из гена bronze 1 маиса проявлял аналогичный эффект в усилении экспрессии. Последовательности интронов традиционным образом встраивали в векторы для трансформации растений, как правило, в пределах нетранслируемой лидерной последовательности.
"Линкер" относится к полинуклеотиду, который содержит последовательность, соединяющую два других полинуклеотида. Длина линкера может составлять по меньшей мере 1, 3, 5, 8, 10, 15, 20, 30, 50, 100, 200, 500, 1000 или 2000 полинуклеотидов. Линкер может быть синтетическим, так что его последовательность не встречается в природе, или он может встречаться в природе, как, например, интрон.
Термин "экзон" относится к части ДНК, которая несет последовательность, кодирующую белок или его часть. Экзоны отделены друг от друга вставочными некодирующими последовательностями (нитронами).
Термин "транзитные пептиды" в целом относится к пептидным молекулам, которые при связывании с представляющим интерес белком направляют белок в конкретную ткань, клетку, местоположение внутри клетки или клеточную органеллу. Примеры включают без ограничения транзитные пептиды хлоропластов, сигнальную последовательность нацеливания в ядро и сигнальные последовательности для вакуолей. Для обеспечения локализации в пластиды можно использовать без ограничения сигнальные пептиды из малой субъединицы рибулозо-бисфосфат-карбоксилазы (Wolter et al. 1988, PNAS 85: 846-850; Nawrath et al., 1994, PNAS 91: 12760-12764), NADP-малатдегидрогеназы (Galiardo et al. 1995, Planta 197: 324-332), глутатион-редуктазы (Creissen et al. 1995, Plant J 8: 167-175) или белка R1 из Lorberth et al. (1998, Nature Biotechnology 16: 473-477).
Используемый в данном документе термин "трансформация" относится к переносу фрагмента нуклеиновой кислоты в геном клетки-хозяина, что приводит к генетически стабильному наследованию. В некоторых конкретных вариантах осуществления введение в растение, часть растения и/или растительную клетку осуществляется путем трансформации, опосредованной бактериями, трансформации путем бомбардировки частицами, трансформации, опосредованной фосфатом кальция, трансформации, опосредованной циклодекстринами, электропорации, трансформации, опосредованной липосомами, трансформации, опосредованной наночастицами, трансформации, опосредованной полимерами, доставки нуклеиновых кислот, опосредованной вирусами, доставки нуклеиновых кислот, опосредованной вискерами, микроинъекции, обработки ультразвуком, инфильтрации, трансформации, опосредованной полиэтиленгликолем, трансформации протопласта или любого другого электрического, химического, физического и/или биологического механизма, который приводит к введению нуклеиновой кислоты в растение, часть растения и/или его клетку, или их комбинации.
Процедуры для трансформации растений хорошо известны и общеприняты в данной области, и они подробно описаны в литературе. Неограничивающие примеры способов трансформации растений включают трансформацию с помощью доставки нуклеиновых кислот, опосредованной бактериями (например, с помощью бактерий из рода Agrobacterium), доставки нуклеиновых кислот, опосредованной вирусами, доставки нуклеиновых кислот, опосредованной вискерами из карбида кремния, доставки нуклеиновых кислот, опосредованной липосомами, микроинъекцию, бомбардировку микрочастицами, трансформацию, опосредованную фосфатом кальция, трансформацию, опосредованную циклодекстринами, электропорацию, трансформацию, опосредованную наночастицами, обработку ультразвуком, инфильтрацию, поглощение нуклеиновых кислот, опосредованное PEG, а также любой другой электрический, химический, физический (механический) и/или биологический механизм, который приводит к введению нуклеиновой кислоты в растительную клетку, включая любую их комбинацию. Общие руководства по разнообразным способам трансформации растений, известным в данной области техники, включают Miki et al. ("Procedures for Introducing Foreign DNA into Plants" в Methods in Plant Molecular Biology and Biotechnology под редакцией Glick, В.R. и Thompson, J.E. (CRC Press, Inc., Boca Raton, 1993), стр. 67-88) и Rakowoczy-Trojanowska (2002, Cell Mol Biol Lett 7:849-858 (2002)).
Таким образом, в некоторых конкретных вариантах осуществления введение в растение, часть растения и/или растительную клетку осуществляют с помощью трансформации, опосредованной бактериями, трансформации путем бомбардировки частицами, трансформации, опосредованной фосфатом кальция, трансформации, опосредованной циклодекстринами, электропорации, трансформации, опосредованной липосомами, трансформации, опосредованной наночастицами, трансформации, опосредованной полимерами, доставки нуклеиновых кислот, опосредованной вирусами, доставки нуклеиновых кислот, опосредованной вискерами, микроинъекции, обработки ультразвуком, инфильтрации, трансформации, опосредованной полиэтиленгликолем, любого другого электрического, химического, физического и/или биологического механизма, который приводит к введению нуклеиновой кислоты в растение, часть растения и/или его клетку, или их комбинации.
Трансформация, опосредованная Agrobacterium, представляет собой способ, широко применяемый для трансформации растений в связи с высокой эффективностью трансформации и в связи с его широкой применимостью в отношении множества различных видов. Трансформация, опосредованная Agrobacterium, как правило, предусматривает перенос бинарного вектора, несущего представляющую интерес чужеродную ДНК, в подходящий штамм Agrobacterium, который может зависеть от дополнительного набора генов vir, которые штамм-хозяин Agrobacterium содержит либо на корезидентной Ti-плазмиде, либо на хромосоме (Uknes et al 1993, Plant Cell 5:159-169). Перенос рекомбинантного бинарного вектора в Agrobacterium можно выполнять с помощью процедуры трехродительского скрещивания с применением Escherichia coli, несущей рекомбинантный бинарный вектор, хелперного штамма Е. coli, несущего плазмиду, которая способна переместить рекомбинантный бинарный вектор в целевой штамм Agrobacterium. В качестве альтернативы рекомбинантный бинарный вектор можно переносить в Agrobacterium путем трансформации нуклеиновой кислотой ( and Willmitzer 1988, Nucleic Acids Res 16:9877).
and Willmitzer 1988, Nucleic Acids Res 16:9877).
Трансформация растения с помощью рекомбинантной Agrobacterium обычно включает совместное культивирование Agrobacterium с эксплантатами из растения, и ее проводят в соответствии со способами, хорошо известными из уровня техники. Трансформированную ткань, которая несет маркер устойчивости к антибиотикам или гербицидам между граничными последовательностями Т-ДНК бинарной плазмиды, обычно регенерируют на селективной среде.
Другой способ трансформации растений, частей растений и растительных клеток включает внедрение инертных или биологически активных частиц в растительные ткани и клетки. См, например, патенты США №№4945050, 5036006 и 5100792. В общем случае этот способ предусматривает внедрение инертных или биологически активных частиц в растительные клетки при условиях, эффективных для проникновения через наружную поверхность клетки и обеспечения встраивания в ее внутреннюю часть. При использовании инертных частиц вектор можно вводить в клетку путем покрытия частиц вектором, содержащим представляющую интерес нуклеиновую кислоту. В качестве альтернативы, клетку или клетки можно окружить вектором таким образом, чтобы вектор переносился в клетку вслед за частицей. Биологически активные частицы (например, высушенные клетки дрожжей, высушенные бактерии или бактериофаг, каждая(-ый) из которых содержит одну или несколько нуклеиновых кислот, предназначенных для введения) также можно внедрять в растительную ткань.
Таким образом, в конкретных вариантах осуществления настоящего изобретения растительную клетку можно трансформировать с помощью любого способа, известного из уровня техники и описываемого в данном документе, и интактные растения можно регенерировать из таких трансформированных клеток с применением любой из множества известных методик. Регенерация растений из растительных клеток, культуры растительных тканей и/или культивируемых протопластов описана, например, в Evans et al. (Handbook of Plant Cell Cultures, Vol. 1, MacMilan Publishing Co. New York (1983)); и Vasil I.R. (ed.) (Cell Culture and Somatic Cell Genetics of Plants, Acad. Press, Orlando, Vol. I (1984), and Vol. II (1986)). Способы отбора трансформированных трансгенных растений, растительных клеток и/или культур растительных тканей являются общепринятыми в данной области, и их можно использовать в способах по настоящему изобретению, предусмотренных в данном документе.
Под терминами "стабильное введение" или "подвергнутый стабильному введению" в контексте полинуклеотида, введенного в клетку, подразумевается, что введенный полинуклеотид стабильно встроен в геном клетки, и, таким образом, клетка является стабильно трансформированной с помощью полинуклеотида.
Используемый в данном документе термин "стабильная трансформация" или "подвергнутый стабильной трансформации" означает, что нуклеиновая кислота введена в клетку и интегрируется в геном клетки. Соответственно, интегрированная нуклеиновая кислота способна наследоваться ее потомками, более конкретно, потомками нескольких последовательных поколений. Используемый в данном документе термин "геном" также включает ядерный и пластидный геном, и, следовательно, предусмотрена интеграция нуклеиновой кислоты, например, в геном хлоропласта. Используемый в данном документе термин "стабильная трансформация" также может относиться к трансгену, который поддерживается внехромосомно, например в виде минихромосомы.
Стабильную трансформацию клетки можно выявлять, например, в анализе с использованием Саузерн-блот-гибридизации геномной ДНК клетки с последовательностями нуклеиновой кислоты, которые специфически гибридизируются с нуклеотидной последовательностью трансгена, введенного в организм (например, растение). Стабильную трансформацию клетки можно выявить, например, в анализе с использованием нозерн-блот-гибридизации РНК клетки с последовательностями нуклеиновой кислоты, которые специфически гибридизируются с нуклеотидной последовательностью трансгена, введенного в растение или другой организм. Стабильную трансформацию клетки также можно выявить, например, с помощью полимеразной цепной реакции (ПЦР) или других реакций амплификации, хорошо известных из уровня техники, в которых используются специфические праймерные последовательности, которые гибридизируются с целевой последовательностью(-ями) трансгена, что приводит к амплификации последовательности трансгена, которую можно выявить в соответствии со стандартными способами. Трансформацию также можно выявить с помощью прямого секвенирования и/или протоколов гибридизации, хорошо известных из уровня техники.
Термин "способ трансформации и регенерации" относится к способу стабильного введения трансгена в растительную клетку и регенерации растения из трансгенной растительной клетки. Используемые в данном документе трансформация и регенерация включают способ отбора, при котором трансген содержит селектируемый маркер, а трансформированная клетка содержит встроенный и экспрессируемый трансген, вследствие чего трансформированная клетка будет выживать и активно развиваться в присутствии средства для отбора. "Регенерация" относится к выращиванию целого растения из растительной клетки, группы растительных клеток или части растения, такой как протопласт, каллюс или часть ткани.
Термины "селектируемый маркер" или "селектируемый маркерный ген" относятся к гену, экспрессия которого в растительной клетке обеспечивает у клетки преимущество при отборе. Термин "положительный отбор" относится к трансформированной клетке, приобретающей способность метаболизировать субстрат, который ранее она не могла использовать или не могла использовать эффективно, как правило, трансформированной и экспрессирующей положительный селектируемый маркерный ген. Эта трансформированная клетка при этом разрастается среди массы из нетрансформированной ткани. Существует множество типов положительного отбора, от неактивных форм регуляторов роста растений, которые затем превращаются в активные формы с помощью перенесенного фермента, до альтернативных источников углеводов, которые не утилизируются эффективно нетрансформированными клетками, например, маннозы, которая в таком случае становится доступной после трансформации геном фермента, например, фосфоманнозоизомеразы, который позволяет ее метаболизировать. Нетрансформированные клетки либо растут медленно по сравнению с трансформированными клетками, либо не растут совсем. Другие типы отбора могут быть обусловлены тем, что клетки, трансформированные селектируемым маркерным геном, приобретают способность к росту в присутствии средства отрицательного отбора, такого как антибиотик или гербицид, в отличие от отсутствия такой способности к росту у нетрансформированных клеток. Преимущество при отборе, которым обладает трансформированная клетка, также может быть обусловлено утратой гена, который имелся ранее, в называемом "отрицательном отборе". При этом добавляют соединение, токсичное только для клеток, которые не утратили специфический ген (отрицательный селектируемый маркерный ген), присутствующий в родительской клетке (как правило, трансген).
Примеры селектируемых маркеров включают без ограничения гены, которые обеспечивают устойчивость или толерантность к антибиотикам, таким как канамицин (Dekeyser et al. 1989, Plant Phys 90: 217-23), спектиномицин (Svab and Maliga 1993, Plant Mol Biol 14: 197-205), стрептомицин (Maliga et al. 1988, Mol Gen Genet 214: 456-459), гигромицин В (Waldron et al. 1985, Plant Mol Biol 5: 103-108), блеомицин (Hille et al. 1986, Plant Mol Biol 7: 171-176), сульфонамиды (Guerineau et al. 1990, Plant Mol Biol 15: 127-136), стрептотрицин (Jelenska et al. 2000, Plant Cell Rep 19: 298-303) или хлорамфеникол (De Block et al. 1984, EMBO J 3: 1681-1689). Другие селектируемые маркеры включают гены, которые обеспечивают устойчивость или толерантность к гербицидам, такие как мутации S4 и/или Hra в ацетолактатсинтазе (ALS), которые придают устойчивость к гербицидам, в том числе сульфонилмочевинам, имидазолинонам, триазолопиримидинам и пиримидинилтиобензоатам; гены 5-енолпирувилшикимат-3-фосфатсинтазы (EPSPS), в том числе без ограничения описываемые в патентах США №№4940935, 5188642, 5633435, 6566587, 7674598 (а также всех родственных заявках), и глифосат-М-ацетилтрансферазы (GAT), которая придает устойчивость к глифосату (Castle et al. 2004, Science 304:1151-1154, и опубликованные патентные заявки США №№20070004912, 20050246798 и 20050060767); BAR, которая придает устойчивость к глюфосинату (см., например, патент США №5561236); арилоксиалканоатдиоксигеназы или AAD-1, AAD-12 или AAD-13, которые придают устойчивость к 2,4-D; гены, такие как HPPD Pseudomonas, которые придают устойчивость к HPPD; мутанты и варианты протофорфириногеноксидазы (РРО), которые придают устойчивость к гербицидам, приводящим к перекисному окислению липидов, в том числе фомесафену, ацифлуорфен-натрию, оксифлуорфену, лактофену, флутиацет-метилу, сафлуфенацилу, флумиоксазину, флумиклорак-пентилу, карфентразон-этилу, сульфентразону; и гены, придающие устойчивость к дикамбе, такие как ген дикамба-монооксигеназы (Herman et al. 2005, J Biol Chem 280: 24759-24767 и патент США №7812224 и родственные заявки и патенты). Другие примеры селектируемых маркеров можно найти в Sundar and Sakthivel (2008, J Plant Physiology 165: 1698-1716), включенный в данный документ посредством ссылки.
Другие системы для отбора включают применение лекарственных препаратов, аналогов метаболитов, промежуточных соединений метаболитов и ферментов для положительного отбора или зависящего от условий положительного отбора трансгенных растений. Примеры включают без ограничения ген, кодирующий фосфоманнозоизомеразу (PMI), при этом средством отбора является манноза, или ген, кодирующий ксилозоизомеразу, при этом средством отбора является D-ксилоза (Haldrup et al. 1998, Plant Mol Biol 37: 287-96). Наконец, в других системах для отбора в качестве средства отбора можно применять не содержащую гормонов среду. Одним неограничивающим примером является гомеозисный ген маиса kn1, эктопическая экспрессия которого приводит к 3-кратному повышению эффективности трансформации (Luo et al. 2006, Plant Cell Rep 25: 403-409). Примеры различных селектируемых маркеров и генов, кодирующих их, раскрыты в Miki and McHugh (J Biotechnol, 2004, 107: 193-232, включенном посредством ссылки).
В некоторых вариантах осуществления настоящего изобретения селектируемый маркер может иметь растительное происхождение. Пример селектируемого маркера, который может иметь растительное происхождение, включает без ограничения 5-енолпирувилшикимат-3-фосфат-синтазу (EPSPS). Фермент 5-енолпирувилшикимат-3-фосфатсинтаза (EPSPS) катализирует ключевую стадию шикиматного пути, общего для биосинтеза ароматических аминокислот у растений. Гербицид глифосат подавляет EPSPS, тем самым уничтожая растение. Трансгенные растения, устойчивые к глифосату, можно получать с помощью введения трансгена модифицированной EPSPS, на который не воздействует глифосат (к примеру, патент США 6040497, включенный посредством ссылки). Другие примеры модифицированной растительной EPSPS, которую можно применять в качестве селектируемого маркера в присутствии глифосата, включают мутант P106L EPSPS риса (Zhou et al 2006, Plant Physiol 140: 184-195) и мутацию P106S в EPSPS подмаренника цепкого (Baerson et al 2002, Plant Physiol 129: 1265-1275). Другие источники EPSPS, которые не имеют растительного происхождения и которые можно применять, чтобы придать толерантность к глифосату, включают без ограничения мутант P101S в EPSPS из Salmonella typhimurium (Comai et al 1985, Nature 317: 741-744) и мутированный вариант CP4 EPSPS из штамма СР4 Agrobacterium sp. (Funke et al 2006, PNAS 103: 13010-13015). Несмотря на то, что ген растительной EPSPS является ядерным, зрелый фермент локализуется в хлоропласте (Mousdale and Coggins 1985, Planta 163:241-249). EPSPS синтезируется в виде белка-предшественника, содержащего транзитный пептид, и затем предшественник транспортируется в строму хлоропласта и подвергается протеолитической обработке с образованием зрелого фермента (della-Cioppa et al. 1986, PNAS 83: 6873-6877). Следовательно, для получения трансгенного растения, которое характеризуется толерантностью к глифосату, можно вводить подходящим образом мутированный вариант EPSPS, который надлежащим образом перемещается в хлоропласт. В этом случае такое трансгенное растения содержит нативный геномный ген EPSPS, а также трансген мутированной EPSPS. В этом случае глифосат можно применять в качестве средства отбора во время проведения способа трансформации и регенерации, при этом выживают лишь те растения или растительная ткань, которые успешно трансформированы трансгеном мутированной EPSPS.
Используемые в данном документе термины "промотор" и "промоторная последовательность" относятся к последовательностям нуклеиновой кислоты, вовлеченным в регуляцию инициации транскрипции. "Растительный промотор" представляет собой промотор, способный инициировать транскрипцию в растительных клетках. Иллюстративные растительные промоторы включают без ограничения получаемые из растений, из вирусов растений и из бактерий, которые содержат гены, экспрессируемые в растительных клетках, таких как Agrobacterium или Rhizobium. "Тканеспецифический промотор" представляет собой промотор, который предпочтительно инициирует транскрипцию в определенной ткани (или комбинации тканей). "Индуцируемый стрессом промотор" представляет собой промотор, который предпочтительно инициирует транскрипцию при определенных условиях окружающей среды (или комбинации условий окружающей среды). "Промотор, специфический в отношении стадии развития " представляет собой промотор, который предпочтительно инициирует транскрипцию в время определенных стадий развития (или комбинации стадий развития).
Используемый в данном документе термин "регуляторные последовательности" относится к нуклеотидным последовательностям, расположенным выше (5'-некодирующие последовательности), в пределах или ниже (3'-некодирующие последовательности) кодирующей последовательности, которые влияют на транскрипцию, процессинг или стабильность РНК, или трансляцию ассоциированной кодирующей последовательности. Регуляторные последовательности включают без ограничения промоторы, энхансеры, экзоны, интроны, трансляционные лидерные последовательности, сигналы терминации и сигнальные последовательности полиаденилирования. Регуляторные последовательности включают природные и синтетические последовательности, а также последовательности, которые могут представлять собой комбинацию синтетических и природных последовательностей. "Энхансер" представляет собой нуклеотидную последовательность, которая может стимулировать активность промотора, и он может представлять собой природный элемент промотора или гетерологичный элемент, введенный для повышения уровня тканевой специфичности промотора. Кодирующая последовательность может находиться на любой нити двухнитевой молекулы ДНК, и она способна функционировать даже в случае ее расположения как выше, так и ниже промотора.
Некоторые варианты осуществления включают сверхэкспрессию одной или нескольких SEQ ID NO: 9-16, и/или снижение экспрессии и/или концентрации (например, уровня) SEQ ID NO: 9-16. В некоторых вариантах осуществления способ и/или композицию по настоящему изобретению можно применять для сверхэкспрессии одной или нескольких SEQ ID NO 9-16, и/или снижения экспрессии и/или концентрации SEQ ID NO: 9-16 тканеспецифическим образом. Например, одна или несколько SEQ ID NO: 9-16 могут быть функционально связаны с последовательностью тканеспецифического промотора для обеспечения тканеспецифической экспрессии (например, экспрессии, специфической в отношении корня и/или зеленой ткани) одной или нескольких SEQ ID NO: 9-16. В некоторых вариантах осуществления обеспечение сверхэкспрессии или тканеспецифической экспрессии одной или нескольких SEQ ID NO: 9-16 может повышать урожайность, повышать стабильность урожайности в условиях стресса, вызванного засухой, и/или повышать устойчивость к стрессу, вызванному засухой, у растения и/или части растения, в которых экспрессируются указанные белки.
В некоторых вариантах осуществления настоящего изобретения предусмотрено растение, имеющее введенный в его геном ген оптимизации потребления воды, где указанный ген оптимизации потребления воды содержит нуклеотидную последовательность, кодирующую по меньшей мере один полипептид, содержащий SEQ ID NO: 9-16.
В некоторых вариантах осуществления указанное растение характеризуется повышенной урожайностью по сравнению с контрольным растением. В некоторых вариантах осуществления повышенная урожайность представляет собой урожайность в условиях дефицита воды.
В некоторых вариантах осуществления родительская линия указанного растения была отобрана или идентифицирована с помощью нуклеотидного зонда или праймера, которые отжигаются с любой из SEQ ID NO: 1-8, и при этом указанная родительская линия обеспечивает повышенную урожайность по сравнению с растением, не содержащим SEQ ID NO: 1-8.
В некоторых вариантах осуществления указанный ген введен путем гетерологичной экспрессии. В некоторых вариантах осуществления указанный ген введен путем редактирования генов. В некоторых вариантах осуществления указанный ген введен путем селекции или интрогрессии признака.
В некоторых вариантах осуществления последовательность нуклеиновой кислоты содержит любую из SEQ ID NO: 1-8.
В некоторых вариантах осуществления повышенная урожайность представляет собой урожайность в условиях дефицита воды.
В некоторых вариантах осуществления указанное растение представляет собой маис.
В некоторых вариантах осуществления указанное растение представляет собой элитного линию или гибрид маиса.
В некоторых вариантах осуществления указанный ген представляет собой нуклеотидную последовательность, характеризующуюся 80-100% гомологией последовательности с любой из SEQ ID NO: 1-8.
В некоторых вариантах осуществления указанное растение также содержит по меньшей мере один из гаплотипов А-М.
В некоторых вариантах осуществления предусмотрены растительная клетка, идиоплазма, пыльца, семя или часть растения от растения по любому из предыдущих вариантов осуществления.
В некоторых вариантах осуществления предусмотрены генотипированное растение, растительная клетка, идиоплазма, пыльца, семя или часть растения, отобранные или идентифицированные на основании выявления любой из SEQ ID NO: 1-8. В некоторых вариантах осуществления настоящего изобретения растение, растительная клетка, идиоплазма, пыльца, семя или часть растения генотипированы путем выделения ДНК из указанного растения, растительной клетки, идиоплазмы, пыльцы, семени или части растения, и ДНК генотипирована с применением либо ПЦР, либо нуклеотидных зондов, которые связываются с любой из SEQ ID NO 1-8.
В другом варианте осуществления предусмотрен способ отбора первого растения или идиоплазмы маиса, которые проявляют либо повышенную урожайность в условиях засухи, либо повышенную урожайность в условиях, отличных от засухи, причем способ предусматривает: а) выделение нуклеиновых кислот из первого растения или идиоплазмы маиса; b) выявление в первом растении или идиоплазме маиса по меньшей мере одного аллеля локуса количественного признака, который ассоциирован с повышенной урожайностью в условиях засухи, где указанный локус количественного признака локализован в хромосомном интервале, фланкированном и включающем маркеры IIM56014 и IIM48939 на хромосоме 1, IIM39140 и IIM40144 на хромосоме 3, IIM6931 и IIM7657 на хромосоме 9, IIM40272 и IIM41535 на хромосоме 2, IIM39102 и IIM40144 на хромосоме 3, IIM25303 и IIM48513 на хромосоме 5, IIM4047 и IIM4978 на хромосоме 9 и IIM19 и IIM818 на хромосоме 10; и с) отбор указанного первого растения или идиоплазмы маиса, или отбор потомков указанного первого растения или идиоплазмы маиса, содержащих по меньшей мере один аллель, ассоциированный с повышенной урожайностью в условиях засухи. Кроме того, предусмотрен способ, где указанный локус количественного признака локализован в хромосомном интервале, фланкированном и включающем IIM56705 и IIM56748 на хромосоме 1; хромосомном интервале, фланкированном и включающем IIM39914 и IIM39941 на хромосоме 3; хромосомном интервале, фланкированном и включающем IIM7249 и IIM7272 на хромосоме 9; хромосомном интервале, фланкированном и включающем IIM40719 и IIM40771 на хромосоме 2; хромосомном интервале, фланкированном и включающем IIM39900 и IIM39935 на хромосоме 3; хромосомном интервале, фланкированном и включающем IIM25799 и IIM25806 на хромосоме 5; хромосомном интервале, фланкированном и включающем IIM4345 и IIM4458 на хромосоме 9; хромосомном интервале, фланкированном и включающем IIM46822 и IIM62316 на хромосоме 10. Способ дополнительно предусматривает скрещивание указанных отобранных первого растения или идиоплазмы маиса со вторым растением или идиоплазмой маиса, и где подвергнутые интрогрессии растение или идиоплазма маиса проявляют повышенную урожайность в условиях засухи. В дополнительном варианте осуществления при этом по меньшей мере один аллель выявляют с применением композиции, содержащей выявляемую метку.
В другом варианте осуществления способ интрогрессии локуса оптимизации потребления воды предусматривает: а) обеспечение первой популяции растений маиса; b) выявление присутствия генетического маркера, который ассоциирован с оптимизацией потребления воды, а также тесно сцеплен и находится в пределах 24 млн. о. от SM2987, в первой популяции; с) отбор одного или нескольких растений с локусом оптимизации потребления воды из первой популяции растений маиса; и d) получение потомства от одного или нескольких растений с локусом оптимизации потребления воды, где потомство проявляет улучшенную оптимизацию потребления воды по сравнению с первой популяцией. В варианте осуществления при этом генетический маркер выявляется в пределах 10 млн. о. от SM2987; 5 млн. о. от SM2987; 1 млн. о. от SM2987; 0,5 млн. п от SM2987. В варианте осуществления при этом выявляемый генетический маркер находится в пределах любого из: хромосомного интервала, содержащего и фланкированного IIM56014 и IIM48939; хромосомного интервала, содержащего и фланкированного IIM59859 и IIM57051; или хромосомного интервала, содержащего и фланкированного IIM56705 и IIM56748. В другом аспекте предусмотрен вариант осуществления, где генетический маркер выбран или тесно ассоциирован с любыми из: IIM56014, IIM56027, IIM56145, IIM56112, IIM56097, IIM56166, IIM56167, IIM56176, IIM56246, IIM56250, IIM56256, IIM56261, IIM56399,IIM59999, IIM59859, IIM59860, IIM56462, IIM56470, IIM56472, IIM56483, IIM56526, IIM56539, IIM56578, IIM56602, IIM56610, IIM56611, IIM61006, IIM56626, IIМ56658, IIM56671, IIM58395, IIM48879, IIM48880, IIM56700, IIM56705, SM2987, IIM56731, IIM56746, IIM56748, IIМ56759, IIM56770, IIM56772, IIM69710, IIM56795 IIM56910, IIM69670, IIM59541, IIM56918, IIM48891, IIM48892, IIM58609, IIM56962, IIM56965, IIM57051, IIM57340, II57586, IIM57589, IIM57605, IIM57609, IIM57611, IIM57612, IIM57620, IIM57626 и IIM48939. Другой аспект представляет собой растение маиса (stiff или non-stiff stalk), полученное за счет этого варианта осуществления.
В другом варианте осуществления способ интрогрессии локуса оптимизации потребления воды предусматривает: а) обеспечение первой популяции растений маиса; b) выявление присутствия генетического маркера, который ассоциирован с оптимизацией потребления воды, а также тесно сцеплен и находится в пределах 10 млн. о. от SM2996, в первой популяции; с) отбор одного или нескольких растений с локусом оптимизации потребления воды из первой популяции растений маиса; и d)
получение потомства от одного или нескольких растений с локусом оптимизации потребления воды, где потомство проявляет улучшенную оптимизацию потребления воды по сравнению с первой популяцией. В дополнительном варианте осуществления при этом выявляемый генетический маркер находится в пределах 0,5 млн. о., 1 млн. о., 2 млн. о. или 5 млн. о. от SM2996. В дополнительном аспекте генетический маркер находится в пределах хромосомного интервала, содержащего любое из следующего: хромосомного интервала, содержащего и фланкированного IIM39140 и IIM40144; хромосомного интервала, содержащего и фланкированного IIM39732 и IIM40055; хромосомного интервала, содержащего и фланкированного IIM39914 и IIM39941. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей IIM39140, IIM39142, IIM39334, IIM39347, IIM39377, IIM39378, IIM39380, IIM39381, IIM39383, IIM39384, IIM39385, IIM39386, IIM39390, IIM39453, IIM39485, IIM39496, IIM39527, IIM39715, IIM39716, IIM39725, IIM39726, IIM39731, IIM39729, IIM39728, IIM39732, IIM39771, IIM39784, IIM39783, IIM39786, IIM39787, IIM39802, IIM39856, IIM39870, IIM39873, IIM39877, IIM39883, IIM39900, IIM39914,, IIM39935, IIM39941, IIM39976, IIM39990, IIM39994, IIM40032, IIM40033, IIM40045, IIM40046, IIM40047, IIM48771, IIM40055, IIM40060, IIM40061, IIM40062, IIM40064, IIM40094, IIM40095, IIM40096, IIM40099, IIM40144 или маркер, тесно сцепленный с любым из приведенных выше. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В дополнительном варианте осуществления предусмотрен способ интрогрессии локуса оптимизации потребления воды, который предусматривает: а) обеспечение первой популяции растений маиса; b) выявление присутствия генетического маркера, который ассоциирован с оптимизацией потребления воды, а также тесно сцеплен и находится в пределах 12 млн. о. от SM2982, в первой популяции; с) отбор одного или нескольких растений с локусом оптимизации потребления воды из первой популяции растений маиса; и d) получение потомства от одного или нескольких растений с локусом оптимизации потребления воды, где потомство проявляет улучшенную оптимизацию потребления воды по сравнению с первой популяцией. В дополнительном аспекте варианта осуществления при этом выявляемый генетический маркер находится в пределах 5 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2982. В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, содержащего любой из хромосомного интервала, определяемого и фланкированного IIM6931 и IIM7657; хромосомного интервала, содержащего и фланкированного IIM7117 и IIM7427; хромосомного интервала, содержащего и фланкированного IIM7204 и IIM7273. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей IIM6931, IIM6934, IIM6946, IIM6961, IIM7041, IIM7054, IIM7055, IIM7086, IIM7101, IIM7104, IIM7105, IIM7109, IIM7110, IIM7114, IIM7117, IIM7141, IIM7151, IIM7151, IIM7163, IIM7168, IIM7166, IIM7178, IIM7184, IIM7183, IIM7204, IIM7231, IIM7235, IIM7249, IIM7272, IIM7273, IIM7275, IIM7284, IIM7283, IIM7285, IIM7318, IIM7319, IIM7345, IIM7351, IIM7354, IIM7384, IIM7386, IIM7388, IIM7397, IIM7417, IIM7427, IIM7463, IIM7480, IIM7481, IIM7548, IIM7613, IIM7616, IIM48034, IIM7636, IIM7653, IIM7657. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В другом варианте осуществления предусмотрен способ интрогрессии локуса оптимизации потребления воды в растение маиса, предусматривающий стадии: а) обеспечения первой популяции растений маиса; b) выявления присутствия генетического маркера, который ассоциирован с оптимизацией потребления воды, а также тесно сцеплен и находится в пределах 10 млн. о. от SM2991, в первой популяции; с) отбора одного или нескольких растений с локусом оптимизации потребления воды из первой популяции растений маиса; и d) получения потомства от одного или нескольких растений с локусом оптимизации потребления воды, где потомство проявляет улучшенную оптимизацию потребления воды по сравнению с первой популяцией. В дополнительном аспекте варианта осуществления при этом выявляемый генетический маркер находится в пределах 5 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2991. В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, выбранного из группы, состоящей из: хромосомного интервала, определяемого и фланкированного IIM40272 и IIM41535; хромосомного интервала, содержащего и фланкированного IIM40486 и IIM40771; хромосомного интервала, содержащего и фланкированного IIM40646 и IIM40768. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей: IIM40272, IIM40279, IIM40301, IIM40310, IIM40311, IIM40440, IIM40442, IIM40463, IIM40486, IIM40522, IIM40627, IIM40646, IIM40709, IIM40719, IIM40768, IIM40771, IIM40775, IIM40788, IIM40789, IIM40790, IIM40795, IIM40802, IIM40804, IIM40837, IIM40839, IIM40848, IIM47120, IIM40862, IIM40863, IIM40888, IIM40893, IIM40909, IIM40928, IIM40931, IIM40932, IIM40940, IIM47155, IIM40936, IIM47156, IIM40991, IIM40998, IIM41001, IIM41008, IIM41013, IIM41033, IIM41064, IIM41153, IIM41229, IIM41230, IIM41247, IIM41259, IIM41261, IIM41263, IIM41283, IIM41287, IIM41310, IIM41321, IIM41359, IIM41357, IIM41366, IIM41377, IIM46720, IIM41412, IIM41430, IIM41448, IIM41456, IIM49103, IIM41479, IIM41509, IIM41535 или маркер, тесно ассоциированный с ними. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В другом варианте осуществления способ интрогрессии локуса оптимизации потребления воды предусматривает стадии: а) обеспечения первой популяции растений маиса; b) выявления присутствия генетического маркера, который ассоциирован с оптимизацией потребления воды, а также тесно сцеплен и находится в пределах 10 млн. о., 5 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2995, в первой популяции; с) отбора одного или нескольких растений с локусом оптимизации потребления воды из первой популяции растений маиса; и d) получения потомства от одного или нескольких растений с локусом оптимизации потребления воды, где потомство проявляет улучшенную оптимизацию потребления воды по сравнению с первой популяцией. В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, выбранного из группы, состоящей из хромосомного интервала, содержащего и фланкированного IIM39102 и IIM40144; хромосомного интервала, содержащего и фланкированного IIM39732 и IIM40064; хромосомного интервала, содержащего и фланкированного IIM39900 и IIM39935. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей: IIM39102, IIM39140, IIM39142, IIM39283, IIM39291, IIM39298, IIM39300, IIM39301, IIM39304, IIM39306, IIM39309, IIM39334, IIM39335, IIM39336, IIM39340, IIM39347, IIM39375, IIM39377, IIM39378, IIM39380, IIM39381, IIM39383, IIM39384, IIM39385, IIM39386, IIM39390, IIM39401, IIM39409, IIM39447, IIM39497, IIM39715, IIM39716, IIM39731, IIM39732, IIM39830, IIM39856, IIM39870, IIM39873, IIM39877, IIM39883, IIM39900, IIM39935, IIM39989, IIM40045, IIM40062, IIM40064, IIM40144 или маркер, тесно ассоциированный с ними. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В другом варианте осуществления способ интрогрессии локуса оптимизации потребления воды в растение маиса предусматривает стадии: а) обеспечения первой популяции растений маиса; b) выявления присутствия генетического маркера, который ассоциирован с оптимизацией потребления воды, а также тесно сцеплен и находится в пределах 20 млн. о., 10 млн. о., 5 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2973, в первой популяции; с) отбора одного или нескольких растений с локусом оптимизации потребления воды из первой популяции растений маиса; и d) получения потомства от одного или нескольких растений с локусом оптимизации потребления воды, где потомство проявляет улучшенную оптимизацию потребления воды по сравнению с первой популяцией. В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, выбранного из группы, состоящей из хромосомного интервала, содержащего и фланкированного IIM25303 и IIM48513; хромосомного интервала, содержащего и фланкированного IIM25545 и IIM25938; хромосомного интервала, содержащего и фланкированного IIM25800 и IIM25805. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей: IIM25303, IIM25304, IIM25320, IIM25350, IIM25391, IIM25399, IIM25400, IIM25402, IIM25407, IIM25414, IIM25429, IIM25442, IIM25449, IIM25526, IIM25543, IIM25545, IIM25600, IIM25688, IIM25694, IIM25731, IIM25740, IIM25799, IIM25800, IIM25805, IIM25806, IIM25819, IIM25820, IIM25821, IIM25823, IIM25824, IIM25828, IIM25830, IIM25856, IIM25864, IIM25870, IIM25895, IIM25905, IIM25921, IIM25938, IIM25939, IIM25945, IIM25965, IIM25966, IIM25968, IIM25975, IIM25978, IIM25983, IIM25984, IIM25987, IIM25999, IIM25999, IIM26009, IIM26023, IIM26084, IIM26119, IIM26132, IIM26133, IIM26145, IIM26151, IIM48428, IIM26170, IIM26175, IIM26226, IIM26263, IIM26264, IIM26267, IIM26268, IIM26271, IIM26272, IIM26273, IIM26274, IIM26291, IIM26319, IIM26323, IIM26325, IIM26383, IIM26402, IIM26493, IIM26495, IIM48513 или маркер, тесно ассоциированный с ними. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В другом варианте осуществления предусмотрен способ интрогрессии локуса оптимизации потребления воды в растение маиса, предусматривающий стадии: a) обеспечения первой популяции растений маиса; b) выявления присутствия генетического маркера, который ассоциирован с оптимизацией потребления воды, а также тесно сцеплен и находится в пределах 10 млн. о., 5 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2980, в первой популяции; с) отбора одного или нескольких растений с локусом оптимизации потребления воды из первой популяции растений маиса; и d) получения потомства от одного или нескольких растений с локусом оптимизации потребления воды, где потомство проявляет улучшенную оптимизацию потребления воды по сравнению с первой популяцией. В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, выбранного из группы, состоящей из: хромосомного интервала, содержащего и фланкированного IIM4047 и IIM4978; хромосомного интервала, содержащего и фланкированного IIM4231 и IIM4607; или хромосомного интервала, содержащего и фланкированного IIM4395 и IIM4458. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей: IIM4047, IIM4046, IIM4044, IIM4038, IIM4109, IIM4121, IIM4143, IIM4177, IIM4203, IIM4212, IIM4214, IIM4214, IIM4215, IIM4219, IIM4226, IIM4227, IIM4229, IIM4231, IIM4232, IIM4233, IIM4235, IIM4236, IIM4237, IIM4239, IIM4239, IIM4240, IIM4241, IIM4242, IIM4244, IIM4255, IIM4263, IIM4264, IIM4265, IIM4308, IIM4295, IIM4289, IIM4280, IIM4345, IIM4387, IIM4387, IIM4388, IIM4388, IIM4389, IIM4390, IIM4390, IIM4392, IIM4395, IIM4458, IIM4469, IIM4482, IIM4607, IIM4608, IIM4609, IIM4613, IIM4614, IIM4674, IIM4681, IIM4682, IIM4738, IIM4755, IIM4756, IIM4768, IIM4777, IIM4816, IIM4818, IIM4822, IIM4831, IIM4851, IIM4856, IIM47276, IIM4857, IIM4858, IIM4859, IIM4860, IIM4875, IIM4878, IIM4967, IIM4974, IIM4978 или маркер, тесно ассоциированный с ними. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В другом варианте осуществления предусмотрен способ интрогрессии локуса оптимизации потребления воды в растение маиса, предусматривающий стадии: а) обеспечения первой популяции растений маиса; b) выявления присутствия генетического маркера, который ассоциирован с оптимизацией потребления воды, а также тесно сцеплен и находится в пределах 5 млн. о., 4 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2984, в первой популяции; с) отбора одного или нескольких растений с локусом оптимизации потребления воды из первой популяции растений маиса; и d) получения потомства от одного или нескольких растений с локусом оптимизации потребления воды, где потомство проявляет улучшенную оптимизацию потребления воды по сравнению с первой популяцией. В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, выбранного из группы, состоящей из хромосомного интервала, содержащего и фланкированного IIM 19 и IIM818; хромосомного интервала, содержащего и фланкированного IIM43 и IIM291, или хромосомного интервала, содержащего и фланкированного IIM121 и IIM211. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей: IIM19, , IIM26, IIM32, IIM43, IIM66, IIM72, IIM78, IIM77, IIM84, IIM108, IIM121, IIM46822, IIM211, IIM236, IIM274, IIM275, IIM291, IIM347, IIM47190, IIM638, IIM738, IIM739, IIM818 или маркер, тесно ассоциированный с ними. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В другом варианте осуществления способ интрогрессии локуса оптимизации потребления воды предусматривает: а) обеспечение первой популяции растений маиса; b) выявление присутствия генетического маркера, который ассоциирован с оптимизацией потребления воды, а также тесно сцеплен и находится в пределах 24 млн. о. от SM2987, в первой популяции; с) отбор одного или нескольких растений с локусом оптимизации потребления воды из первой популяции растений маиса; и d) получение потомства от одного или нескольких растений с локусом оптимизации потребления воды, где потомство проявляет улучшенную оптимизацию потребления воды по сравнению с первой популяцией. В варианте осуществления при этом генетический маркер выявляется в пределах 10 млн. о. от SM2987; 5 млн. о. от SM2987; 1 млн. о. от SM2987; 0,5 млн. п от SM2987. В варианте осуществления при этом выявляемый генетический маркер находится в пределах любого из: хромосомного интервала, содержащего и фланкированного IIM56014 и IIM48939; хромосомного интервала, содержащего и фланкированного IIM59859 и IIM57051; или хромосомного интервала, содержащего и фланкированного IIM56705 и IIM56748. В другом аспекте предусмотрен вариант осуществления, где генетический маркер выбран или тесно ассоциирован с любыми из: IIM56014, IIM56027, IIM56145, IIM56112, IIM56097, IIM56166, IIM56167, IIM56176, IIM56246, IIM56250, IIM56256, IIM56261, IIM56399, IIM59999, IIM59859, IIM59860, IIM56462, IIM56470, IIM56472, IIM56483, IIM56526, IIM56539, IIM56578, IIM56602, IIM56610, IIM56611, IIM61006, IIM56626, IIM56658, IIM56671, IIM58395, IIM48879, IIM48880, IIM56700, IIM56705, SM2987, IIM56731, IIM56746, IIM56748, IIM56759, IIM56770, IIM56772, IIM69710, IIM56795 IIM56910, IIM69670, IIM59541, IIM56918, IIM48891, IIM48892, IIM58609, IIM56962, IIM56965, IIM57051, IIM57340, IIM57586, IIM57589, IIM57605, IIM57609, IIM57611, IIM57612, IIM57620, IIM57626 и IIM48939. Другой аспект представляет собой растение маиса (stiff или non-stiff stalk), полученное за счет этого варианта осуществления.
В другом варианте осуществления способ интрогрессии локуса оптимизации потребления воды предусматривает: а) обеспечение первой популяции растений маиса; Ь) выявление присутствия генетического маркера, который ассоциирован с оптимизацией потребления воды, а также тесно сцеплен и находится в пределах 10 млн. о. от SM2996, в первой популяции; с) отбор одного или нескольких растений с локусом оптимизации потребления воды из первой популяции растений маиса; и d) получение потомства от одного или нескольких растений с локусом оптимизации потребления воды, где потомство проявляет улучшенную оптимизацию потребления воды по сравнению с первой популяцией. В дополнительном варианте осуществления при этом выявляемый генетический маркер находится в пределах 0,5 млн. о., 1 млн. о., 2 млн. о. или 5 млн. о. от SM2996. В дополнительном аспекте генетический маркер находится в пределах хромосомного интервала, содержащего любое из следующего: хромосомного интервала, содержащего и фланкированного IIM39140 и IIM40144; хромосомного интервала, содержащего и фланкированного IIM39732 и IIM40055; или хромосомного интервала, содержащего и фланкированного IIM39914 и IIM39941. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей IIM39140, IIM39142, IIM39334, IIM39347, IIM39377, IIM39378, IIM39380, IIM39381, IIM39383, IIM39384, IIM39385, IIM39386, IIM39390, IIM39453, IIM39485, IIM39496, IIM39527, IIM39715, IIM39716, IIM39725, IIM39726, IIM39731, IIM39729, IIM39728, IIM39732, IIM39771, IIM39784, IIM39783, IIM39786, IIM39787, IIM39802, IIM39856, IIM39870, IIM39873, IIM39877, IIM39883, IIM39900, IIM39914,, IIM39935, IIM39941, IIM39976, IIM39990, IIM39994, IIM40032, IIM40033, IIM40045, IIM40046, IIM40047, IIM48771, IIM40055, IIM40060, IIM40061, IIM40062, IIM40064, IIM40094, IIM40095, IIM40096, IIM40099, IIM40144 или маркер, тесно сцепленный с любым из приведенных выше. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В дополнительном варианте осуществления предусмотрен способ интрогрессии локуса оптимизации потребления воды, который предусматривает: а) обеспечение первой популяции растений маиса; b) выявление присутствия генетического маркера, который ассоциирован с оптимизацией потребления воды, а также тесно сцеплен и находится в пределах 12 млн. о. от SM2982, в первой популяции; с) отбор одного или нескольких растений с локусом оптимизации потребления воды из первой популяции растений маиса; и d) получение потомства от одного или нескольких растений с локусом оптимизации потребления воды, где потомство проявляет улучшенную оптимизацию потребления воды по сравнению с первой популяцией. В дополнительном аспекте варианта осуществления при этом выявляемый генетический маркер находится в пределах 5 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2982. В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, содержащего любой из хромосомного интервала, определяемого и фланкированного IIM6931 и IIM7657; хромосомного интервала, содержащего и фланкированного IIM7117 и IIM7427; хромосомного интервала, содержащего и фланкированного IIM7204 и IIM7273. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей IIM6931IIM6934, IIM6946, IIM6961, IIM7041, IIM7054, IIM7055, IIM7086, IIM7101, IIM7104, IIM7105, IIM7109, IIM7110, IIM7114, IIM7117, IIM7141, IIM7151, IIM7151, IIM7163, IIM7168, IIM7166, IIM7178, IIM7184, IIM7183, IIM7204, IIM7231, IIM7235, IIM7249, IIM7272, IIM7273, IIM7275, IIM7284, IIM7283, IIM7285, IIM7318, IIM7319, IIM7345, IIM7351, IIM7354, IIM7384, IIM7386, IIM7388, IIM7397, IIM7417, IIM7427, IIM7463, IIM7480, IIM7481, IIM7548, IIM7613, IIM7616, IIM48034, IIM7636, IIM7653, IIM7657. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В другом варианте осуществления предусмотрен способ идентификации или отбора растения маиса, характеризующегося повышенной урожайностью в условиях засухи или повышенной урожайностью в условиях, отличных от засухи, по сравнению с контрольным растением, где урожайность представляет собой увеличение количества бушелей кукурузы на акр, причем способ предусматривает стадии: а) выделения нуклеиновой кислоты из растительной клетки; b) выявления в указанной нуклеиновой кислоте присутствия генетического маркера, который ассоциирован с повышенной урожайностью (в условиях засухи или условиях, отличных от засухи), где указанный генетический маркер тесно сцеплен и находится в пределах 10 млн. о., 5 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2991; с) отбора растения маиса на основе генетического маркера, выявленного на b). В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, выбранного из группы, состоящей из: хромосомного интервала, определяемого и фланкированного IIM40272 и IIM41535; хромосомного интервала, содержащего и фланкированного IIM40486 и IIM40771; хромосомного интервала, содержащего и фланкированного IIM40646 и IIM40768. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей: IIM40272, IIM40279, IIM40301, IIM40310, IIM40311, IIM40440, IIM40442, IIM40463, IIM40486, IIM40522, IIM40627, IIM40646, IIM40709, IIM40719, IIM40768, IIM40771, IIM40775, IIM40788, IIM40789, IIM40790, IIM40795, IIM40802, IIM40804, IIM40837, IIM40839, IIM40848, IIM47120, IIM40862, IIM40863, IIM40888, IIM40893, IIM40909, IIM40928, IIM40931, IIM40932, IIM40940, IIM47155, IIM40936, IIM47156, IIM40991, IIM40998, IIM41001, IIM41008, IIM41013, IIM41033, IIM41064, IIM41153, IIM41229, IIM41230, IIM41247, IIM41259, IIM41261, IIM41263, IIM41283, IIM41287, IIM41310, IIM41321, IIM41359, IIM41357, IIM41366, IIM41377, IIM46720, IIM41412, IIM41430, IIM41448, IIM41456, IIM49103, IIM41479, IIM41509, IIM41535 или маркер, тесно ассоциированный с ними. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), отобранные с помощью способа выше.
В другом варианте осуществления предусмотрен способ идентификации или отбора растения маиса, характеризующегося повышенной урожайностью в условиях засухи или повышенной урожайностью в условиях, отличных от засухи, по сравнению с контрольным растением, где урожайность представляет собой увеличение количества бушелей кукурузы на акр, причем способ предусматривает стадии: а) выделения нуклеиновой кислоты из растительной клетки; b) выявления в указанной нуклеиновой кислоте присутствия генетического маркера, который ассоциирован с повышенной урожайностью (в условиях засухи или условиях, отличных от засухи), где указанный генетический маркер тесно сцеплен и находится в пределах 10 млн. о., 5 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2995; с) отбора растения маиса на основе генетического маркера, выявленного на b). В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, выбранного из группы, состоящей из хромосомного интервала, содержащего и фланкированного IIM39102 и IIM40144; хромосомного интервала, содержащего и фланкированного IIM39732 и IIM40064; хромосомного интервала, содержащего и фланкированного IIM39900 и IIM39935. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей: IIM39102, IIM39140, IIM39142, IIM39283, IIM39291, IIM39298, IIM39300, IIM39301, IIM39304, IIM39306, IIM39309, IIM39334, IIM39335, IIM39336, IIM39340, IIM39347, IIM39375, IIM39377, IIM39378, IM39380, IIM39381, IIM39383, IIM39384, IIM39385, IIM39386, IIM39390, IIM39401, IIM39409, IIM39447, IIM39497, IIM39715, IIM39716, IIM39731, IIM39732, IIM39830, IIM39856, IIM39870, IIM39873, IIM39877, IIM39883, IIM39900, IIM39935, IIM39989, IIM40045, IIM40062, IIM40064, IIM40144 или маркер, тесно ассоциированный с ними. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), отобранные с помощью способа выше.
В другом варианте осуществления предусмотрен способ идентификации или отбора растения маиса, характеризующегося повышенной урожайностью в условиях засухи или повышенной урожайностью в условиях, отличных от засухи, по сравнению с контрольным растением, где урожайность представляет собой увеличение количества бушелей кукурузы на акр, причем способ предусматривает стадии: а) выделения нуклеиновой кислоты из растительной клетки; b) выявления в указанной нуклеиновой кислоте присутствия генетического маркера, который ассоциирован с повышенной урожайностью (в условиях засухи или в условиях, отличных от засухи), где указанный генетический маркер тесно сцеплен и находится в пределах 20 млн. о., 10 млн. о., 5 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2973; с) отбора растения маиса на основе генетического маркера, выявленного на b). В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, выбранного из группы, состоящей из хромосомного интервала, содержащего и фланкированного IIM25303 и IIM48513; хромосомного интервала, содержащего и фланкированного IIM25545 и IIM25938; хромосомного интервала, содержащего и фланкированного IIM25800 и IIM25805. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей: IIM25303, IIM25304, IIM25320, IIM25350, IIM25391, IIM25399, IIM25400, IIM25402, IIM25407, IIM25414, IIM25429, IIM25442, IIM25449, IIM25526, IIM25543, IIM25545, IIM25600, IIM25688, IIM25694, IIM25731, IIM25740, IIM25799, IIM25800, IIM25805, IIM25806, IIM25819, IIM25820, IIM25821, IIM25823, IIM25824, IIM25828, IIM25830, IIM25856, IIM25864, IIM25870, IIM25895, IIM25905, IIM25921, IIM25938, IIM25939, IIM25945, IIM25965, IIM25966, IIM25968, IIM25975, IIM25978, IIM25983, IIM25984, IIM25987, IIM25999, IIM25999, IIM26009, IIM26023, IIM26084, IIM26119, IIM26132, IIM26133, IIM26145, IIM26151, IIM48428, IIM26170, IIM26175, IIM26226, IIM26263, IIM26264, IIM26267, IIM26268, IIM26271, IIM26272, IIM26273, IIM26274, IIM26291, IIM26319, IIM26323, IIM26325, IIM26383, IIM26402, IIM26493, IIM26495, IIM48513 или маркер, тесно ассоциированный с ними. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В другом варианте осуществления предусмотрен способ идентификации или отбора растения маиса, характеризующегося повышенной урожайностью в условиях засухи или повышенной урожайностью в условиях, отличных от засухи, по сравнению с контрольным растением, где урожайность представляет собой увеличение количества бушелей кукурузы на акр, причем способ предусматривает стадии: а) выделения нуклеиновой кислоты из растительной клетки; b) выявления в указанной нуклеиновой кислоте присутствия генетического маркера, который ассоциирован с повышенной урожайностью (в условиях засухи или в условиях, отличных от засухи), где указанный генетический маркер тесно сцеплен и находится в пределах 10 млн. о., 5 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2980; с) отбора растения маиса на основе генетического маркера, выявленного на b). В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, выбранного из группы, состоящей из: хромосомного интервала, содержащего и фланкированного IIM4047 и IIM4978; хромосомного интервала, содержащего и фланкированного IIM4231 и IIM4607; или хромосомного интервала, содержащего и фланкированного ПМ4395 и IIM4458. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей: IIM4047, IIM4046, IIM4044, IIM4038, IIM4109, IIM4121, IIM4143, IIM4177, IIM4203, IIM4212, IIM4214, IIM4214, IIM4215, IIM4219, IIM4226, IIM4227, IIM4229, IIM4231, IIM4232, IIM4233, IIM4235, IIM4236, IIM4237, IIM4239, IIM4239, IIM4240, IIM4241, IIM4242, IIM4244, IIM4255, IIM4263, IIM4264, IIM4265, IIM4308, IIM4295, IIM4289, IIM4280, IIM4345, IIM4387, IIM4387, IIM4388, IIM4388, IIM4389, IIM4390, IIM4390, IIM4392, IIM4395, IIM4458, IIM4469, IIM4482, IIM4607, IIM4608, IIM4609, IIM4613, IIM4614, IIM4674, IIM4681, IIM4682, IIM4738, IIM4755, IIM4756, IIM4768, IIM4777, IIM4816, IIM4818, IIM4822, IIM4831, IIM4851, IIM4856, IIM47276, IIM4857, IIM4858, IIM4859, IIM4860, IIM4875, IIM4878, IIM4967, IIM4974, IIM4978 или маркер, тесно ассоциированный с ними. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В другом варианте осуществления предусмотрен способ идентификации или отбора растения маиса, характеризующегося повышенной урожайностью в условиях засухи или повышенной урожайностью в условиях, отличных от засухи, по сравнению с контрольным растением, где урожайность представляет собой увеличение количества бушелей кукурузы на акр, причем способ предусматривает стадии: а) выделения нуклеиновой кислоты из растительной клетки; b) выявления в указанной нуклеиновой кислоте присутствия генетического маркера, который ассоциирован с повышенной урожайностью (в условиях засухи или в условиях, отличных от засухи), где указанный генетический маркер тесно сцеплен и находится в пределах 5 млн. о., 4 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. о. от SM2984; с) отбора растения маиса на основе генетического маркера, выявленного на b). В другом аспекте при этом выявляемый генетический маркер находится в пределах хромосомного интервала, выбранного из группы, состоящей из хромосомного интервала, содержащего и фланкированного IIM 19 и IIM818; хромосомного интервала, содержащего и фланкированного IIM43 и IIM291, или хромосомного интервала, содержащего и фланкированного IIM121 и IIM211. В другом аспекте варианта осуществления выявляемый генетический маркер выбран из группы, содержащей: IIM19, IIM26, IIM32, IIM43, IIM66, IIM72, IIM78, IIM77, IIM84, IIM108, IIM121, IIM46822, IIM211, IIM236, IIM274, IIM275, IIM291, IIM347, IIM47190, IIM638, IIM738, IIM739, IIM818 или маркер, тесно ассоциированный с ними. Дополнительный аспект варианта осуществления представляет собой клетку растения маиса или растение маиса (stiff или non-stiff stalk), полученные с помощью способа выше.
В другом варианте осуществления предусмотрен способ получения гибридного растения с повышенной урожайностью в условиях засухи или в условиях, отличных от засухи, по сравнению с контролем, который предусматривает стадии: (а) обеспечения первого растения, содержащего первый генотип, содержащий любой из гаплотипов А-М; (b) обеспечения второго растения, содержащего второй генотип, содержащий любой маркер из группы, включающей SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 или SM2984, где второе растение содержит по меньшей мере один маркер из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 или SM2984, который не присутствует в первом растении; (с) скрещивания первого растения и второго растения маиса с получением поколения F1; при этом с идентификацией одного или нескольких представителей поколения F1, которые содержат требуемый генотип, содержащий любую комбинацию из гаплотипов А-М и любых маркеров из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 или SM2984, где требуемый генотип отличается как от первого генотипа из (а), так и от второго генотипа из (b), с получением тем самым гибридного растения с повышенной оптимизацией потребления воды. В дополнительном варианте осуществления при этом гибридное растение с повышенной урожайностью содержит каждый из гаплотипов А-М, которые присутствуют в первом растении, а также по меньшей мере один дополнительный гаплотип, выбранный из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 или SM2984, который присутствует во втором растении. В дополнительном аспекте варианта осуществления при этом первое растение представляет собой реккурентного родителя, содержащего по меньшей мере один из гаплотипов А-М, а второе растение представляет собой донора, который содержит по меньшей мере один маркер из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 или SM2984, который не присутствует в первом растении. В другом аспекте варианта осуществления при этом первое растение является гомозиготным по меньшей мере по двум, трем, четырем или пяти из гаплотипов А-М. В другом аспекте гибридное растение содержит по меньшей мере три, четыре, пять, шесть, семь, восемь или девять из гаплотипов А-М и маркеры из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 или SM2984. В дополнительном аспекте при этом идентификация предусматривает генотипирование одного или нескольких представителей поколения F1, полученного за счет скрещивания первого растения и второго растения, в отношении каждого из гаплотипов А-М и маркеров из группы, состоящей из SM2987, SM2991, SM2995, SM2996, SM2973, SM2980, SM2982 и SM2984, присутствующих либо в первом растении, либо во втором растении. В дополнительном аспекте при этом первое растение и второе растение представляют собой растения Zea mays. Предусмотрен вариант осуществления, где повышенная урожайность представляет собой Т повышенную или стабилизованную урожайность в обстановке недостатка воды по сравнению с контрольным растением. В дополнительном аспекте при этом гибрид с повышенной урожайностью можно высаживать при большей густоте посева и/или обеспечивается отсутствие груза для урожая, когда он находится при благоприятных уровнях обеспеченности влагой. Другой аспект представляет собой гибридное растение Zea mays, полученное с помощью варианта осуществления, или его клетку, культуру тканей, семя или часть.
Другой вариант осуществления настоящего изобретения представляет собой растение, имеющее введенный в его геном ген оптимизации потребления воды, где указанный ген оптимизации потребления воды содержит нуклеотидную последовательность, кодирующую по меньшей мере один полипептид, содержащий SEQ ID NO: 9-16, и дополнительно, где введение указанного гена оптимизации потребления воды повышает урожайность в условиях засухи или в условиях, отличных от засухи. В другом аспекте варианта осуществления при этом введение представляет собой любое из интрогрессии посредством селекции растений, редактирования генома (TALEN, CRISPR и т.д.) или трансгенной экспрессии. В другом аспекте варианта осуществления при этом указанное растение характеризуется повышенной урожайностью по сравнению с контрольным растением. В другом аспекте при этом повышенная урожайность представляет собой урожайность в условиях дефицита воды. В дополнительном аспекте при этом родительская линия указанного растения была выбрана или идентифицирована с помощью нуклеотидного зонда или праймера, которые отжигаются с любой из SEQ ID NO: 1-8, и при этом указанная родительская линия обеспечивает повышенную урожайность по сравнению с растением, не содержащим SEQ ID NO: 1-8. В другом аспекте при этом повышенная урожайность растения представляет собой урожайность в условиях достаточного количества воды. В дополнительном аспекте при этом растение представляет собой маис, гибридное растение маиса или элитную линию маиса. В дополнительном аспекте при этом указанный ген представляет собой нуклеотидную последовательность, характеризующуюся 90-100% гомологией последовательности с любой из SEQ ID NO: 1-8. В дополнительном аспекте варианта осуществления при этом указанное растение также содержит по меньшей мере один из гаплотипов А-М.
Другой вариант осуществления предусматривает генотипированное растение, растительную клетку, идиоплазму, пыльцу, семя или часть растения, выбранные или идентифицированные на основании выявления любой из SEQ ID NO: 1-8 или маркеров, тесно ассоциированных с ними (например, показанных в таблицах 1-7). В дополнительном аспекте варианта осуществления при этом растение, растительная клетка, идиоплазма, пыльца, семя или часть растения генотипированы путем выделения ДНК из указанного растения, растительной клетки, идиоплазмы, пыльцы, семени или части растения, и ДНК генотипирована с применением либо ПЦР, либо нуклеотидных зондов, которые связываются с любой из SEQ ID NO 1-8.
Другой вариант осуществления представляет собой способ получения растения с повышенной урожайностью, предусматривающий стадии: а) отбора из разнородной популяции растений с применением маркера, выбранного из группы, состоящей из маркеров SM2973, SM2980, SM2982, SM2984, SM2987, SM2991, SM2995, SM2996; b) размножения/самоопыления растения. В другом аспекте маркер SM2973 имеет "G" в нуклеотиде 401; маркер SM2980 имеет "С" в нуклеотиде 401; маркер SM2982 имеет "А" в нуклеотиде 401; маркер SM2984 имеет "G" в нуклеотиде 401; маркер SM2987 имеет "G" в нуклеотиде 401; маркер SM2991 имеет "G" в нуклеотиде 401; маркер SM2995 имеет "А" в нуклеотиде 401; и маркер SM2996 имеет "А" в нуклеотиде 401.
В другом варианте осуществления предусмотрен способ идентификации или отбора растения маиса, характеризующегося повышенной урожайностью в условиях засухи или повышенной урожайностью в условиях, отличных от засухи, по сравнению с контрольным растением, где урожайность представляет собой увеличение количества бушелей кукурузы на акр, причем способ предусматривает стадии: а) выделения нуклеиновой кислоты из растительной клетки; b) выявления в указанной нуклеиновой кислоте присутствия генетического маркера, который ассоциирован с повышенной урожайностью (в условиях засухи или в условиях, отличных от засухи), где указанный генетический маркер тесно сцеплен и находится в пределах 10 млн. о., 5 млн. о., 2 млн. о., 1 млн. о. или 0,5 млн. п. от гена маиса, выбранного из группы, состоящей из GRMZM5G862107_01; GRMZM2G094428_01; GRMZM2G027059_01; GRMZM2G050774_01; GRMZM2G134234_03; GRMZM2G416751_02; GRMZM2G467169_02; GRMZM2G156365_06 или любой их комбинации; и с) отбора растения маиса на основании генетического маркера, выявленного на b).
В другом варианте осуществления культурное растение содержит в пределах своего генома кассету для экспрессии в растении, где указанная кассета для экспрессии содержит растительный промотор (конститутивный, или ткане/клетка-специфический или предпочтительный), функционально связанный с геном, содержащим последовательность ДНК, характеризующуюся 70%, 80%, 90%, 95%, 99% или 100% идентичностью последовательности с любой из SEQ ID NO: 1-8, где термин "культурное растение" в данном документе означает однодольные растения, такие как зерновые культуры (пшеница, просо, сорго, рожь, тритикале, разновидности овса, ячмень, абиссинская трава, спельта, гречиха, голодный рис и киноа), рис, маис (кукуруза) и/или сахарный тростник; и/или двудольные сельскохозяйственные культуры, такие как свекла (такая как сахарная свекла или кормовая свекла), плодовые растения (такие как семечковые плодовые растения, косточковые плодовые растения или ягодные растения, например, разновидности яблони, груши, сливы, персика, миндаля, вишни, клубники, малины или ежевики); бобовые растения (такие как разновидности фасоли, чечевицы, гороха или сои); масличные растения (такие как рапс, горчица, мак, разновидности маслины, разновидности подсолнечника, кокосовая пальма, растения клещевины, какао-бобы или разновидности арахиса), огуречные растения (такие как разновидности кабачка, огурца или дыни); волокнистые растения (такие как хлопчатник, лен, конопля или джут), цитрусовые плодовые растения (такие как разновидности апельсина, лимона, грейпфрута или мандарина), овощные растения (такие как шпинат, салат-латук, разновидности кочанной капусты, моркови, томата, картофеля, тыквы или стручковый перец); лавровые (такие как разновидности авокадо, коричное дерево или камфорное дерево); табак; ореховые растения; кофейное дерево; чайный куст; разновидности винограда; разновидности хмеля; дуриан; разновидности банана; каучуковые растения и декоративные растения (такие как цветочные растения, кустарники, широколиственные деревья или вечнозеленые растения, например, хвойные). Данный перечень не является каким-либо ограничением.
В другом варианте осуществления культурное растение содержит в пределах своего генома кассету для экспрессии в растении, где указанная кассета для экспрессии содержит растительный промотор (конститутивный, или ткане/клетка-специфический или предпочтительный), функционально связанный с геном, кодирующим белок, характеризующийся 70%, 80%, 90%, 95%, 99% или 100% идентичностью последовательности с любой из SEQ ID NO: 9-16.
В другом варианте осуществления предусмотрен способ получения растения маиса, характеризующегося повышенной урожайностью в условиях засухи или повышенной урожайностью в условиях, отличных от засухи, где повышенная урожайность представляет собой увеличение количества бушелей на акр по сравнению с контрольным растением, причем способ предусматривает стадии: (а) выделения нуклеиновой кислоты из растительной клетки; (b) редактирования геномной последовательности растительной клетки из а), чтобы она содержала молекулярный маркер, ассоциированный с повышенной засухоустойчивостью, где молекулярный маркер представляет собой любой молекулярный маркер, описанный в таблицах 1-7, и дополнительно, где геномная последовательность не имела указанный молекулярный маркер ранее; и (с) получения растения или растительного каллюса из растительной клетки из (b). В другом аспекте варианта осуществления можно получать матрицу на основе нуклеиновой кислоты для облегчения получения отредактированной последовательности(-ей), где специалист в данной области смог бы применять известные инструменты для редактирования генома для прямого получения отредактированных последовательностей в пределах генома целевого растения (например, редактирование генома проводили с помощью способов редактирования генома CRISPR, TALEN или мегануклеаз, известных из уровня техники). В другом аспекте варианта осуществления при этом отредактированная последовательность содержит любое из следующего, соответствующее:
i. SM2987, расположенному на хромосоме 1 маиса, который соответствует аллелю G в положении 272937870;
ii. SM2991, расположенному на хромосоме 2 маиса, который соответствует аллелю G в положении 12023706;
iii. SM2995, расположенному на хромосоме 3 маиса, который соответствует аллелю А в положении 225037602;
iv. SM2996, расположенному на хромосоме 3 маиса, который соответствует аллелю А в положении 225340931;
v. SM2973, расположенному на хромосоме 5 маиса, который соответствует аллелю G в положении 159121201; (6)
vi. SM2980, расположенному на хромосоме 9 маиса, который соответствует аллелю С в положении 12104936;
vii. SM2982, расположенному на хромосоме 9 маиса, который соответствует аллелю А в положении 133887717; или
viii. SM2984, расположенному на хромосоме 10 маиса, который соответствует аллелю G в положении 4987333; и
Не ограничиваясь теорией, в другом варианте осуществления растения по настоящему изобретению содержат улучшенные агрономические признаки, такие как мощность проростков, потенциально возможная урожайность, поглощение фосфатов, рост растения, рост проростков, поглощение фосфора, устойчивость к полеганию, развитие генеративных органов или качество зерна.
В другом варианте осуществления охватывается применение молекулярного маркера в пределах хромосомного интервала для отбора, идентификации или получения растения маиса, характеризующегося повышенной засухоустойчивостью и/или урожайность, где хромосомный интервал представляет собой любое из: интервала, расположенного в пределах 20 сМ, 15 сМ, 10 сМ, 9 сМ, 8 сМ, 7 сМ, 6 сМ, 5 сМ, 4 сМ, 3 сМ, 2 сМ, 1 сМ, или тесно сцепленного с аллелем урожайности, соответствующим любому из: SM2987, расположенному на хромосоме 1 маиса, соответствующему аллелю G в положении 272937870; SM2991, расположенному на хромосоме 2 маиса, соответствующему аллелю G в положении 12023706; SM2995, расположенному на хромосоме маиса 3, соответствующему аллелю А, в положении 225037602; SM2996, расположенному на хромосоме 3 маиса, соответствующему аллелю А в положении 225340931; SM2973, расположенному на хромосоме 5 маиса, соответствующему аллелю G в положении 159121201; SM2980, расположенному на хромосоме 9 маиса, соответствующему аллелю С в положении 12104936; SM2982, расположенному на хромосоме 9 маиса, соответствующему аллелю А в положении 133887717; или SM2984, расположенному на хромосоме 10 маиса, соответствующему аллелю G в положении 4987333; или
хромосомного интервала, фланкированного и включающего любой из: IIM56014 и IIM48939 на хромосоме 1 маиса, расположенных в физических положениях пар оснований 248150852-296905665, IIM39140 и IIM40144 на хромосоме 3 маиса, расположенных в физических положениях пар оснований 201538048-230992107, IIM6931 и IIM7657 на хромосоме 9 маис, расположенных в физических положениях пар оснований 121587239-145891243, IIM40272 и IIM41535 на хромосоме 2 маиса, расположенных в физических положениях пар оснований 1317414-36929703, IIM25303 и IIM48513 на хромосоме 5 маиса, расположенных в физических положениях пар оснований 139231600-183321037, IIM4047 и IIM4978 на хромосоме 9 маиса, расположенных в физических положениях пар оснований 405220-34086738, или IIM19 и IIM818 на хромосоме 10 маиса, расположенных в физических положениях пар оснований 1285447-29536061.
В другом варианте осуществления применение любого аллеля, перечисленного в таблицах 1-7, служит для получения отредактированных последовательностей генома, или модификацию осуществляют для получения растения с повышенной урожайностью в условиях засухи и/или условиях, отличных от засухи.
Таким образом, в некоторых вариантах осуществления объектов, раскрытых в настоящем изобретении, предусмотрены инбредные растения Zea mays, содержащие один или несколько аллелей, ассоциированных с повышенной урожайностью, повышенной урожайностью в условиях засухи или требуемым признаком оптимизации потребления воды.
ПРИМЕРЫ
В следующих примерах представлены иллюстративные варианты осуществления. В свете настоящего изобретения и общего уровня знаний в данной области специалисты в данной области будут понимать, что следующие примеры предусмотрены только в качестве иллюстрации, и что можно использовать многочисленные замены, модификации и изменения без отклонения от объема объектов, раскрытых в настоящем изобретении.
Введение в ПРИМЕРЫ
Для оценки значения различных молекулярных маркеров/аллелей в условиях стресса, вызванного засухой, разнородную идиоплазму подвергали скринингу в условиях контролируемых полевых экспериментов, предусматривающих контрольную обработку с обильным поливом и обработку с ограниченным поливом. Цель обработки с обильным поливом состоит в том, чтобы убедиться, что вода не ограничивает продуктивность сельскохозяйственной культуры. Напротив, цель обработки с ограниченным поливом состоит в том, чтобы убедиться, что вода является главным лимитирующим ограничением для урожайности зерна. Основные эффекты (например, обработка и генотип) и взаимодействия (например, генотип х обработка) можно определять, когда две обработки применяют рядом друг с другом в полевых условиях. Помимо этого, можно оценивать количественно фенотипы, связанные с засухой, для каждого генотипа в панели, обеспечивая тем самым возможность проводить ассоциации маркер-признак.
При осуществлении на практике способ обработки с ограниченным поливом можно варьировать в широких пределах, в зависимости от идиоплазмы, подлежащей скринингу, типа почвы, климатических условий в месте произрастания, снабжения водой до сезона вегетации и снабжения водой во время сезона вегетации, при этом упомянуты только некоторые. Сперва идентифицируют место произрастания, в котором количество осадков во время сезона вегетации является низким (для сведения к минимуму вероятности непредусмотренной подачи воды) и которое подходит для культивирования растений. Кроме того, может быть важно определить временные рамки стресса, вследствие чего параметр определяют так, чтобы убедиться, что имеет место согласованность скрининга в разные годы и в разных местоположениях. Также следует учитывать понимание интенсивности обработки или, в некоторых случаях, потери урожая, ожидаемые при обработке с ограниченным поливом. Выбор интенсивности обработки, которая является слишком слабой, может не позволить выявить генотипическую изменчивость. Выбор интенсивности обработки, которая является слишком сильной, может приводить к большой экспериментальной ошибке. После того как идентифицированы временные рамки стресса и описана интенсивность обработки, полив можно контролировать способом, который согласуется с этими параметрами.
Общие способы оценки и оценки засухоустойчивости можно найти в Salekdeh et al., 2009 и в патенте США №№: 6635803; 7314757; 7332651 и 7432416.
Пример 1. Идентификация генетических локусов маиса, ассоциированных с урожайностью в условиях засухи и условиях, отличных от засухи
Анализ с полногеномным поиском ассоциаций (GWA) проводили путем тестирования однонуклеотидных полиморфизмов (SNP) в генах в отношении ассоциации с признаками, связанными с засухой, измеренными в маисе с помощью панели ассоциации с оптимизацией потребления воды (WO). В данной работе идентифицировали локусы, маркеры, аллели и QTL, ассоциированные с признаками урожайности в условиях засухи или в условиях достаточного количества воды.
Генотипирование и обнаружение маркеров
Примерно 1,09 миллиона маркеров SNP идентифицировали среди 754 разнородных линий маиса с применением технологии секвенирования нового поколения. Чтобы экстраполировать полногеномное покрытие маркеров на основании этого набора данных, 21,8 миллиона маркеров, опубликованных в НарМар2 для маиса (Chia et al. Nat. Gen. 2012 44:803-809), повторно картировали на сборке В73 RefGen_v2 (www.геном.arizona.edu/modules/publisher/item.php?itemid=16). Перекрывание 26 родительских линий NAM (Buckler et al. Science 2009 325:714-718) применяли для подстановки маркеров Panzea НарМар2 по всей панели. Чтобы уменьшить ошибки генотипирования, пороговое значение эмпирически полученной предсказанной ошибки (расчетная процентная доля неправильно подставленных генотипов), составляющее 0,025, применяли для фильтрации 21,8 миллиона маркеров до 9,7 миллиона маркеров для дальнейшего анализа. Данные маркеры подвергали дополнительной фильтрации, учитывая в первой фазе анализа только генные маркеры SNP, что привело к 1,4 миллиона SNP. Примером подходящего способа подстановки является пакет программного обеспечения NPUTE (Roberts et al. Bioinformatics 2007 23:i401-i407).
Фенотипические данные
Из 754 линий маиса, проанализированных в отношении данных о маркерах SNP, для 512 линий получили данные по урожайности, доступные из предшествующих испытаний в условиях засухи. Измеряли два признака урожайности для оценки засухоустойчивости, в частности, урожайности в условиях полива (YGSMN_i) или урожайности в условиях стресса, вызванного засухой (YGSMN_s). Показатели для каждой линии измерял в разных условиях окружающей среды. Наилучшие линейные прогнозы (BLUP), рассчитанные для переменных условий окружающей среды, коррелировали с YGSMN_i и YGSMN_s (r = 0,63, Р < 0,001). Все анализы ассоциации проводили с такими BLUP для каждого признака отдельно. Фенотипические данные и генотипические данные маиса объединяли для идентификации хромосомных интервалов, QTL и SNP, имеющих значимую ассоциацию с урожайностью в условиях засухи и условиях, отличных от засухи.
Анализ ассоциаций
Из 1,4 миллиона генных маркеров SNP примерно 780000 SNP исходно проверяли на ассоциацию с данными по урожайности. Остальные 620000 маркеров были мономорфными у всех 512 линий с данными по урожайности и, следовательно, не обладали мощностью для анализа ассоциаций в отношении урожайности в условиях засухи и условиях, отличных от засухи. Остальные 780000 SNP разбивали на наборы из 10000 смежных маркеров и тестировали в анализе ассоциаций с данными по урожайности с применением унифицированной смешанной модели (Zhang et al. Nat. Gen. 2010 42:355-362). Три разные унифицированные смешанные модели тестировали со всеми данными в следующем формате:
у = Pv + Sa + Iu + е,
где y представляет собой вектор для фенотипических значений, v представляет собой вектор фиксированных эффектов, относящихся к структуре популяции, α представляет собой фиксированный эффект кандидатного маркера, u представляет собой вектор для случайных эффектов, связанных с последними общими предками, а е представляет собой вектор для остатков. P представляет собой матрицу векторов, определяющих структуру популяции, S представляет собой вектор для генотипов по кандидатному маркеру, а I представляет собой матрицу идентичности. Считается, что дисперсия случайных эффектов представляет собой Var(u) = 2KVg и Var(e) = IVR, где K представляет собой матрицу родства, состоящую из относительного числа значений для общих аллелей, и I представляет собой матрицу идентичности.
Три смешанные модели тестировали, чтобы оценить три различные способа для расчета матрицы родства и для определения того, следует ли принадлежность к группам селекции включать как фиксированные эффекты в модели. Для первой модели (под названием модель QLocalK) Р определяли как принадлежность к семи из девяти групп селекции. Лишь восемь из девяти групп селекции были представлены в нашей панели, что привело к включению семи векторов (восьмой был необязательным, поскольку компоненты вектора для каждой особи в сумме составляли единицу). Для каждого набора из 10000 смежных маркеров уникальную матрицу родства рассчитывали и включали в модель. Аналогично во второй модели (под названием модель QGlobalK) Р определяли как принадлежность к семи из девяти групп селекции. Однако вместо матрицы локального родства, рассчитанной на основе набора из 10000 смежных маркеров, рассчитывали матрицу глобального родства, исходя из 10000 маркеров, выбранных случайным образом из генома. Эту матрицу глобального родства применяли для тестирования всех маркеров. И наконец, тестировали третью модель (под названием модель ChrK), которая не включала фиксированный эффект структуры популяции (без условия Р), а просто матрицу хромосомного родства. В данной модели применяли матрицы родства, специфичные для каждой хромосомы, исходя из данных микрочипа из 55 тыс. маркеров из MaizeSNP50 BeadChip (Illumina, Сан Диего, Калифорния). Эти матрицы родства включали информацию для 478 линий с фенотипическими данными по урожайности в условиях полива и для 479 линий с данными по урожайности в условиях стресса. Каждый маркер затем тестировали с соответствующей хромосомной матрицей K. Все ассоциации создавали с применением Tassel версии 3.0 (август 2012) (Bradbury et al. Bioinformatics 2007 23:2633-2635), с применением как ранее определенных параметров популяции (P3D), так и сжатых MLM (Zhang et al. Nat. Gen. 2010 42:355-362).
Пошаговая регрессия
Среди SNP, которые оказались значимо ассоциироваными с урожайностью в условиях стресса, только те SNP, которые наблюдали по меньшей мере у 20 из 512 линий с фенотипическими данными, учитывали при создании моделей пошаговой регрессии, чтобы убедиться в применимости найденных маркеров для разнородной популяции маиса. Пошаговую регрессию проводили с применением SAS-процедуры GLMSelect. GLMSelect обеспечивает возможность конкурентной реализации прямого отбора и обратного исключения, исходя из скорректированного R2 для моделей. Оптимизацию модели останавливают, как только получают специфическую предсказанную остаточную сумму квадратов для модели. В пределах гетерозисной группы структуру учитывал путем введения принадлежности к группе селекции в качестве фиксированного эффекта.
SNP, ассоциированные с урожайностью в условиях полива и стресса
Как указано выше, три разных модели, контролирующие структуру популяции разными способами, применяли для тестирования ассоциации всех 780000 SNP как с урожайностью в условиях стресса (YGSMN_s), так и с урожайностью в условиях полива (YGSMN_i), измеряемой во всех местоположениях.
В общей сложности, ровно 771698 SNP тестировали на ассоциацию с урожайностью в условиях полива (YGSMN_i), измеряемую в нескольких местоположениях. Затем ассоциацию с маркерами, при которой минорный аллель наблюдали только у трех особей или меньше, отфильтровывали, что привело к тестированию 262081 SNP. Среди протестированных 427 SNP были значимо ассоциированы (Р < 0,001) с урожайностью в условиях полива.
Немного больше SNP (772008) тестировали на ассоциацию с урожайностью в условиях стресса (YGSMN_s), измеряемую во всех местоположениях. И снова маркеры, где минорный аллель наблюдали только у трех особе или меньше, отфильтровывали, что привело к тестировали 262224 SNP. Однако меньшее количество (268) было значимо ассоциировано (Р < 0,001) с данным признаком по сравнению с урожайностью в условиях полива. И снова, шесть SNP оставались значимо ассоциированными с YGSMN_s, в случае применения порогового значения Р < 10-5. Аналогично тому, что наблюдалось для YGSMN_i, LD быстро падало у SNP, значимо ассоциированных с YGSMN_s, с идентификацией тем самым нескольких потенциальных причинных SNP и/или генов.
Исходя из анализа ассоциаций, несколько генов были идентифицированы как прочно ассоциированные с повышенной урожайностью в условиях, отличных от засухи, и повышенной урожайностью в условиях стресса, вызванного засухой, при этом они включают: GRMZM2G027059, GRMZM2G156365, GRMZM2G134234, GRMZM2G094428, GRMZM2G416751, GRMZM2G467169, GRMZM5G862107 и GRMZM2G050774. Кроме того, картировали маркеры, тесно ассоциированные с данными соответствующими генами, и они аналогичным образом были ассоциированы с повышенной урожайностью в условиях засухи и условиях, отличных от засухи (полный перечень см. в таблицах 1-7; также в таблицах 10а и 10b и таблице 11 показаны результаты картирования ассоциаций для инбредных линий маиса).
Таблица 10а и 10b. Примеры аллелей маиса, которые ассоциированы с урожайностью у различных гетерозисных групп маиса Эффект измеряли в YGSMN_i и YGSMN_s. Во всех случаях показано повышение количества бушелей на акр в условиях засухи и условиях, отличных от засухи, у линий маиса как non-stiff stalk (NSS), так и stiff stalk (SS) по сравнению с контролями.
*Статистические показатели, специфичны для таких SNP в пределах модели пошаговой регрессии.
§В пределах гетерозисной группы размеры эффекта рассчитывали для каждого маркера отдельно.
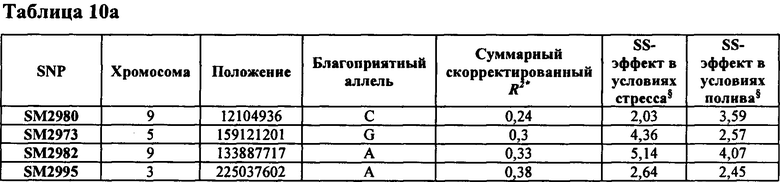
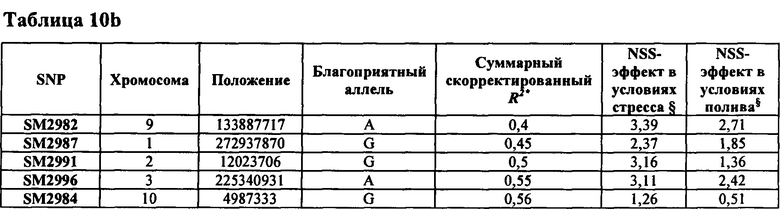
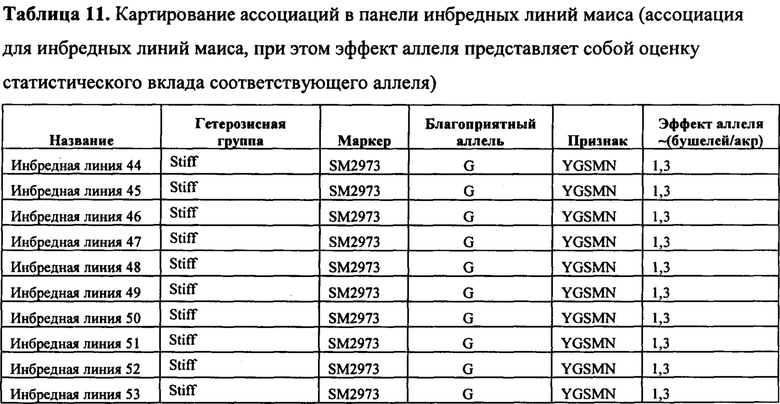
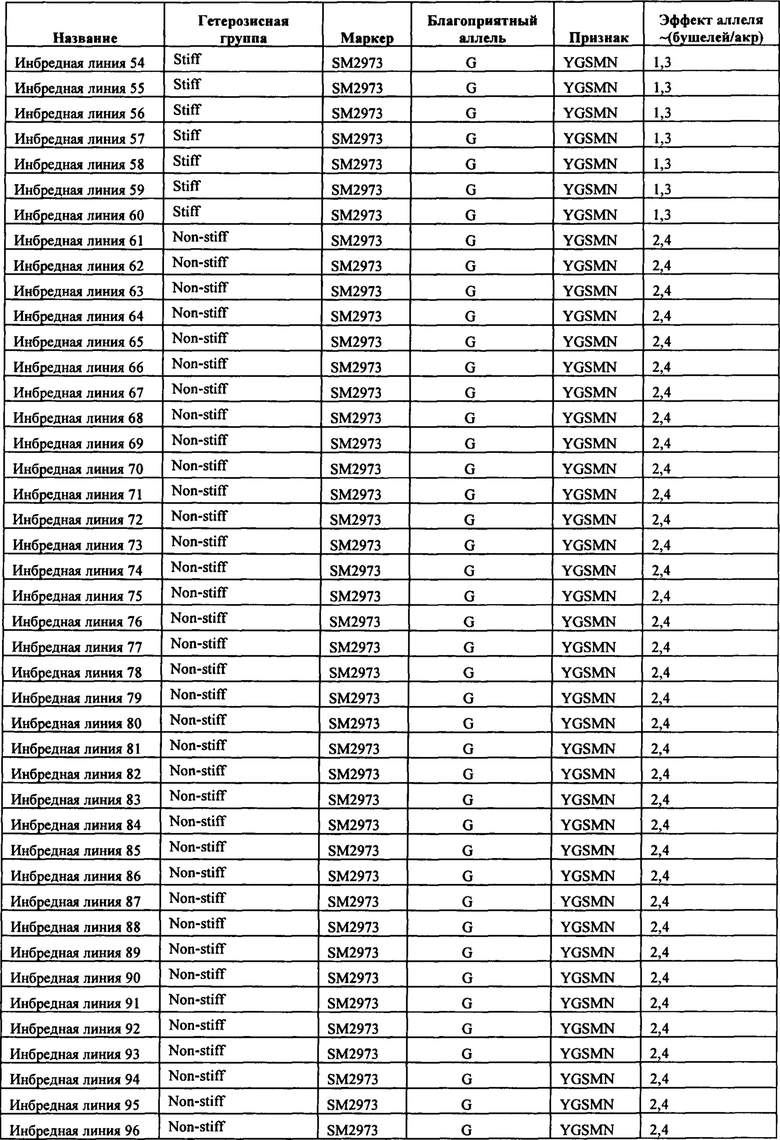
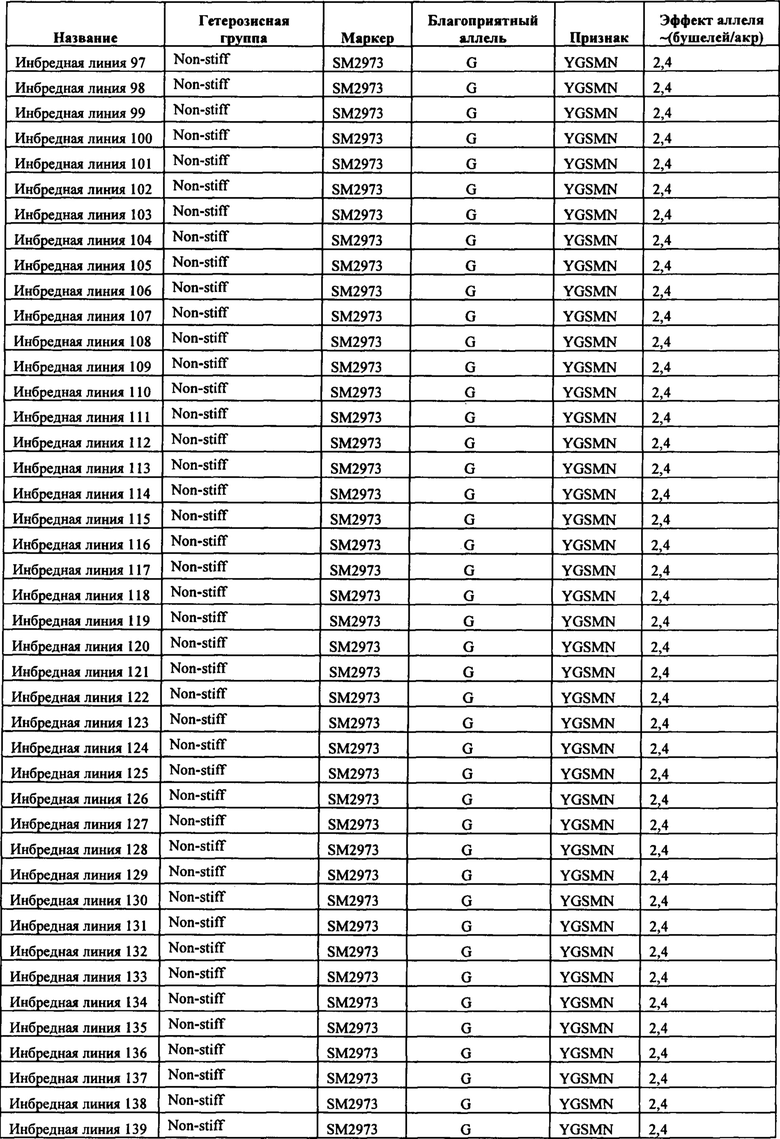
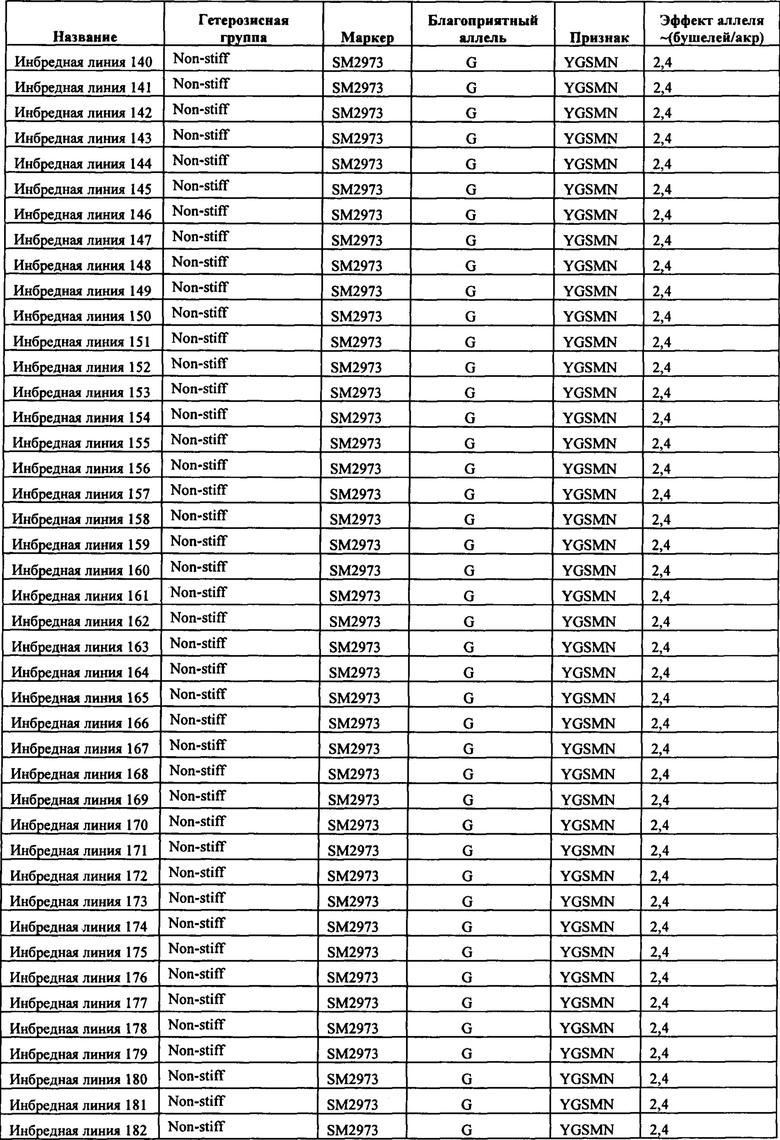
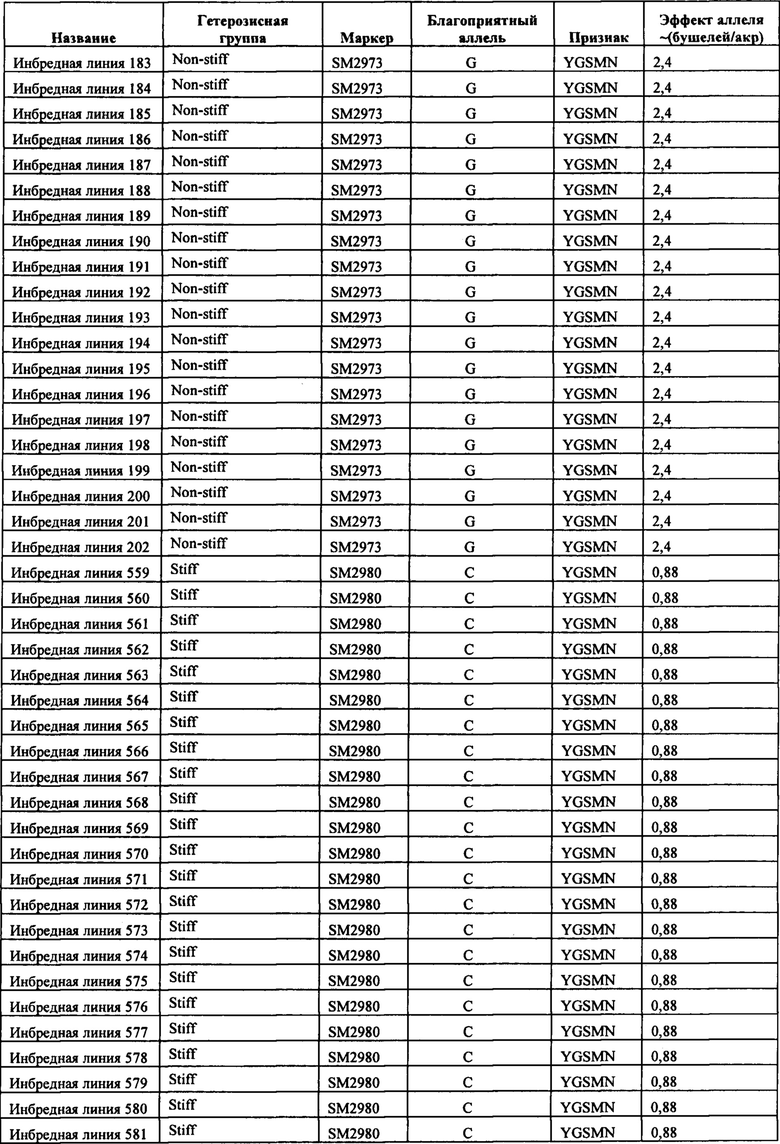
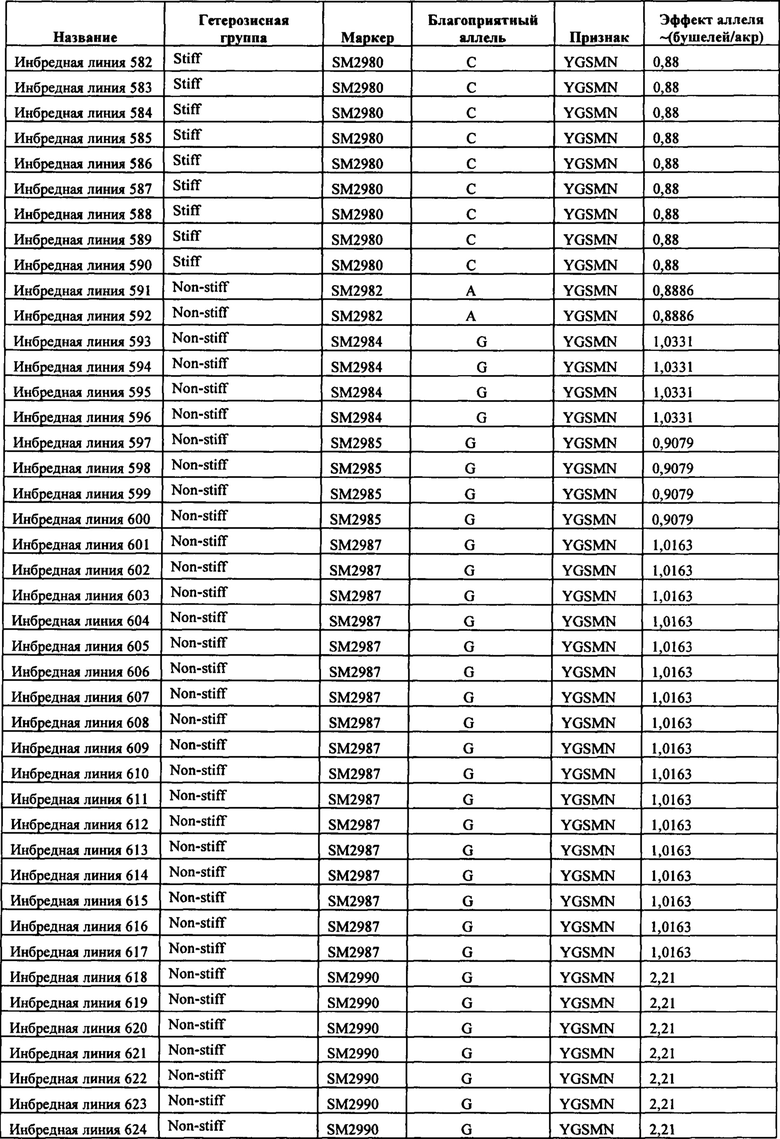
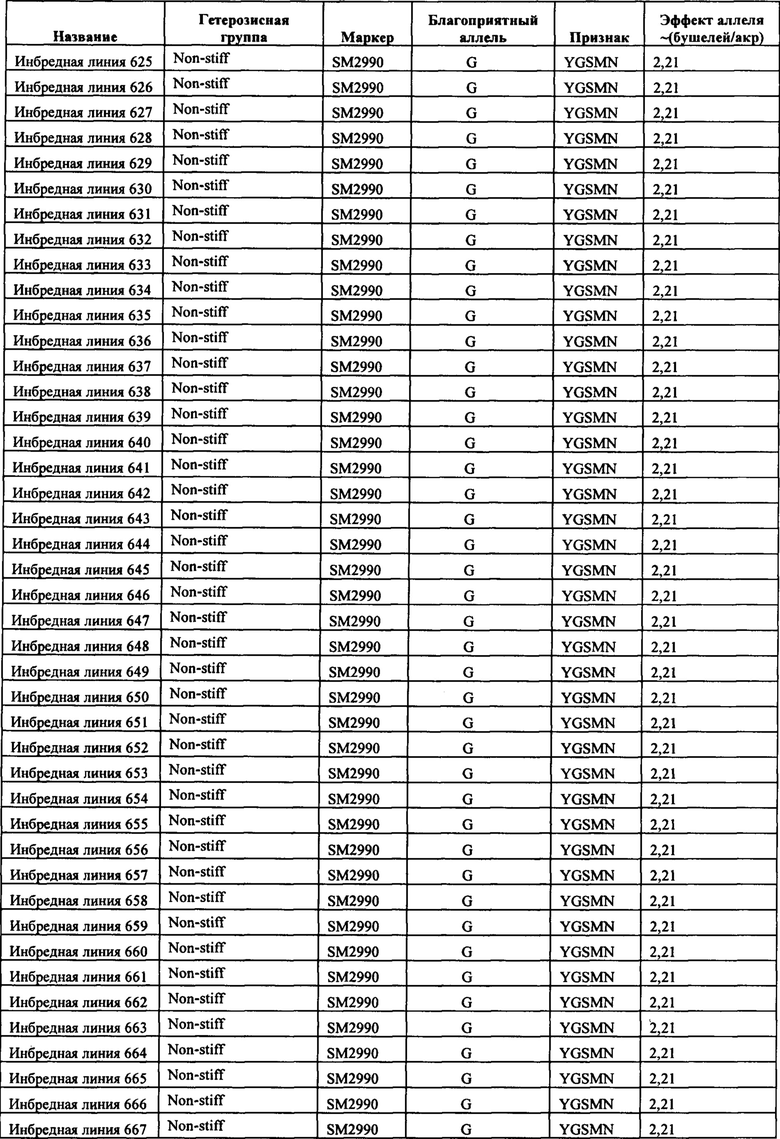
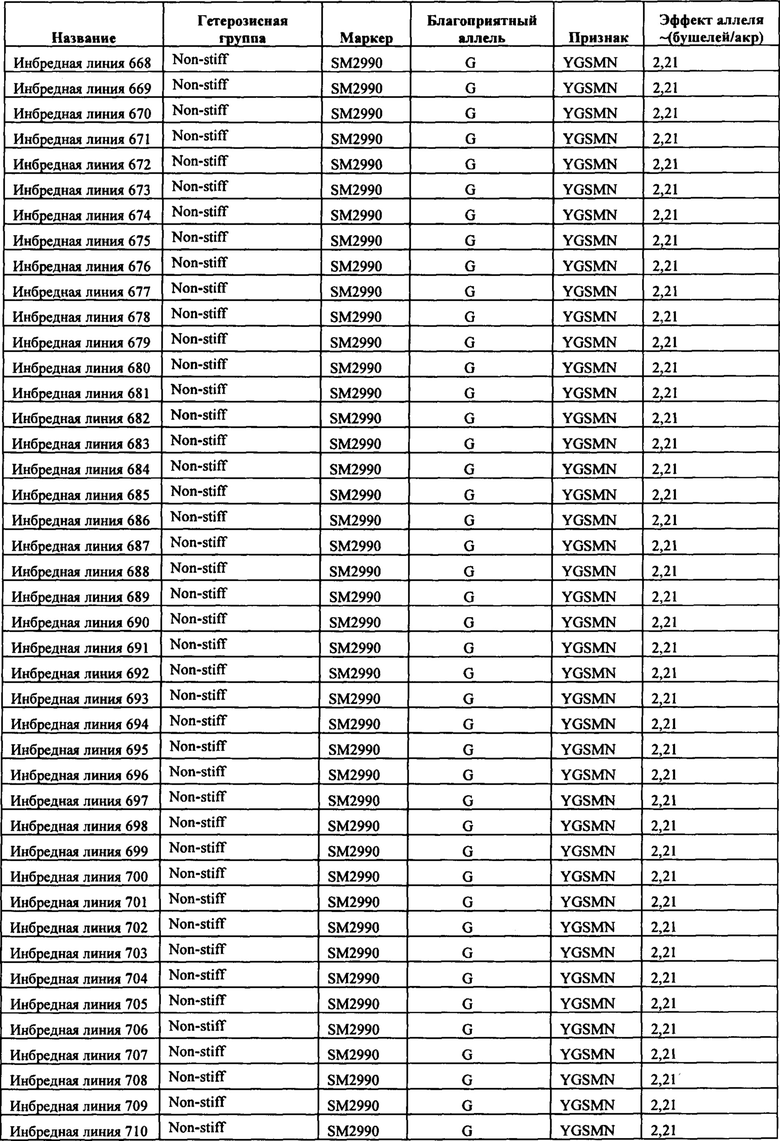
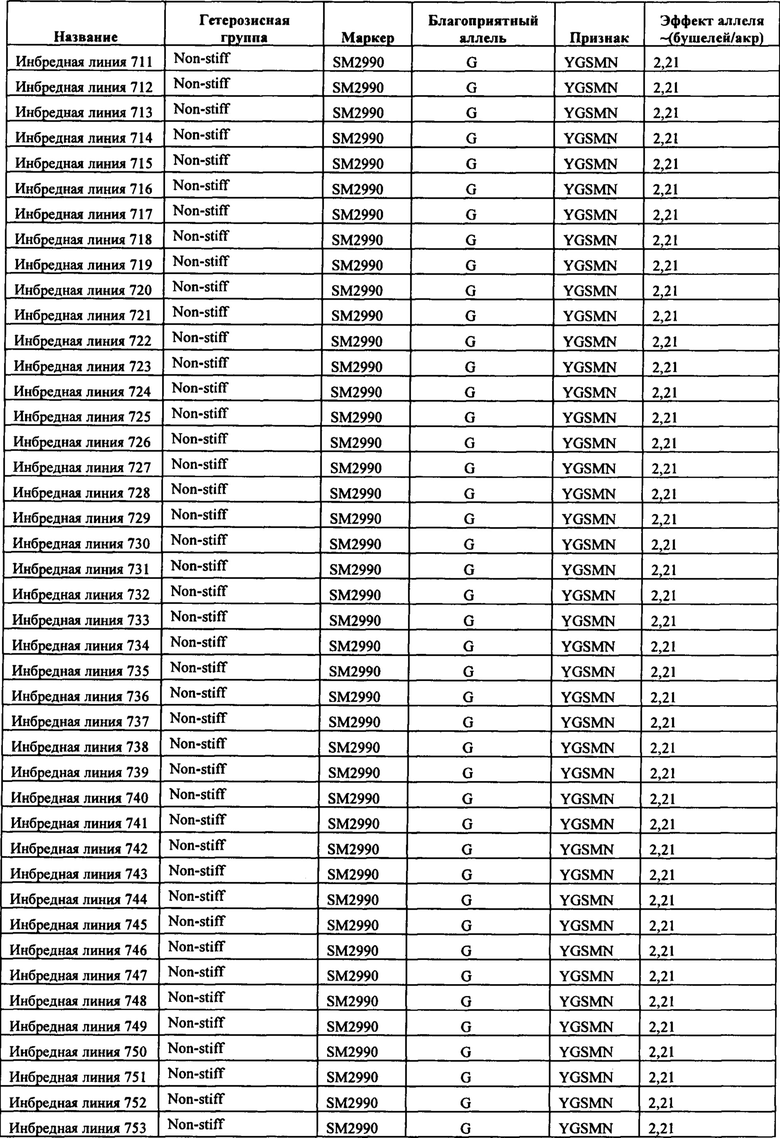
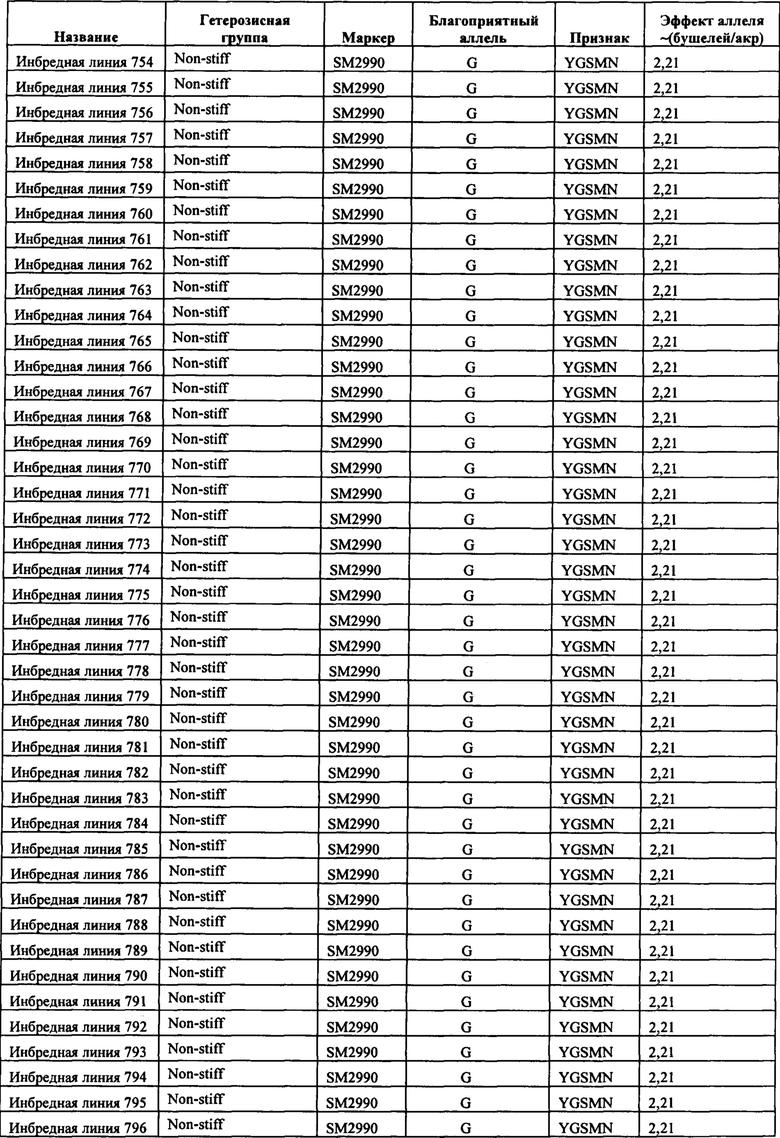
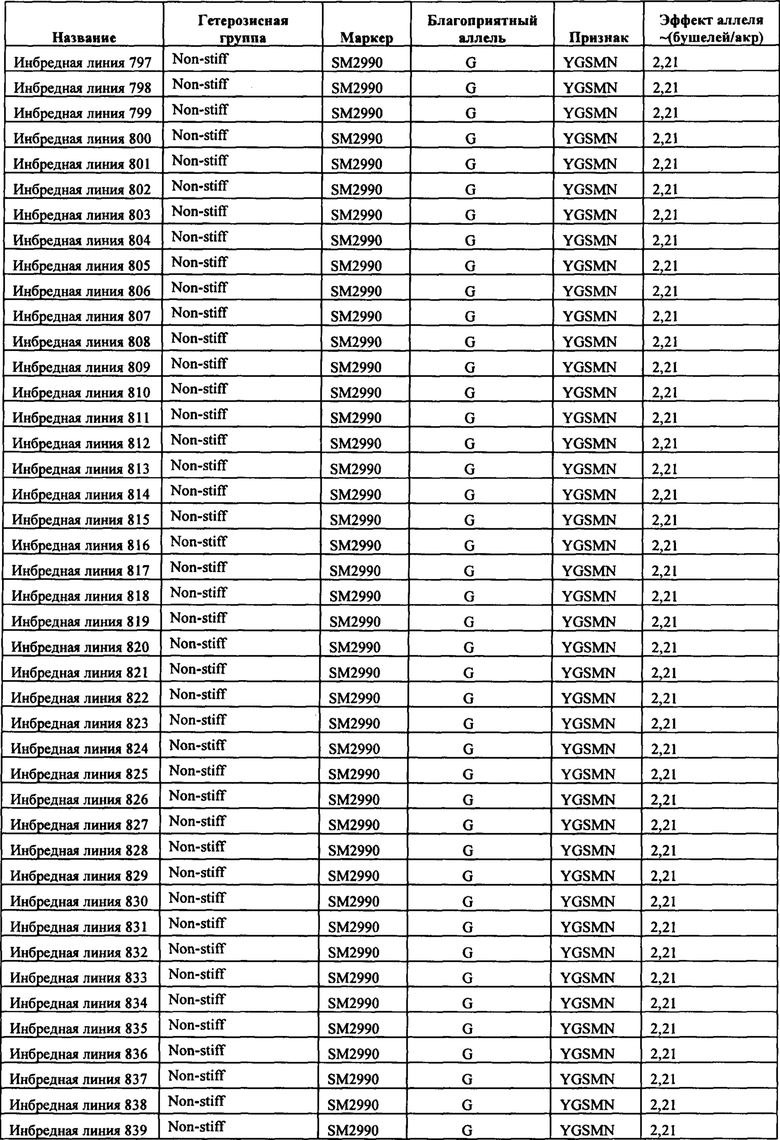
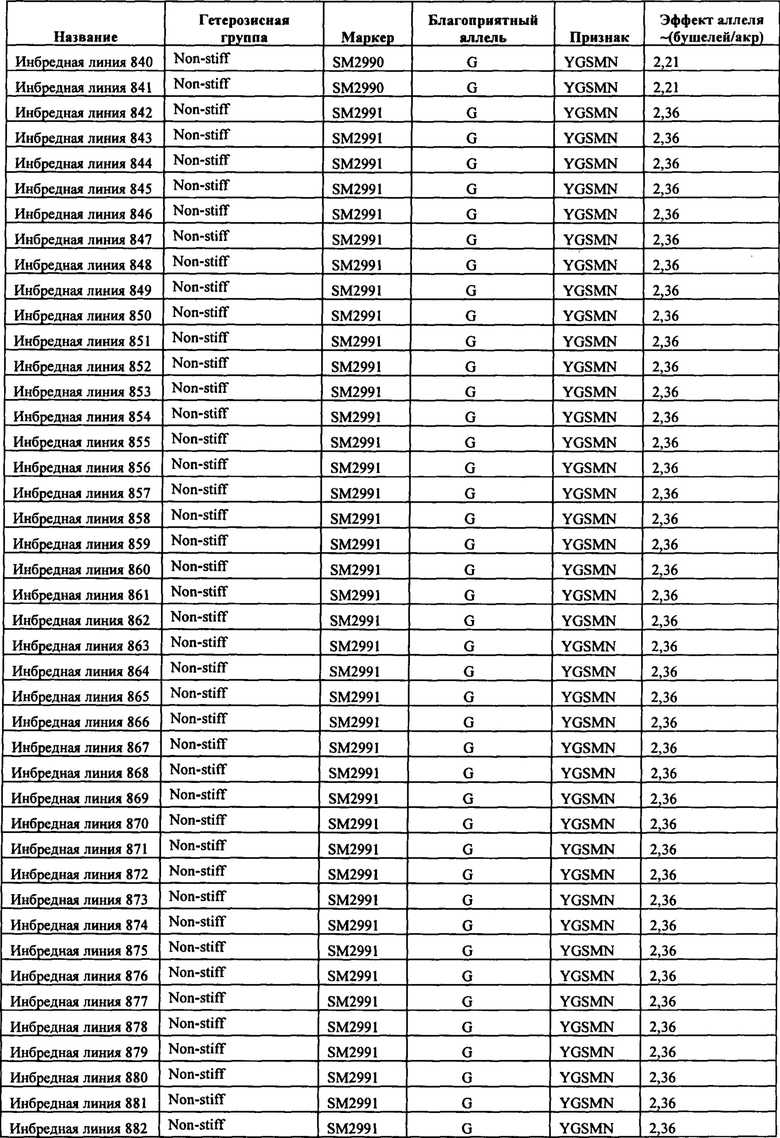
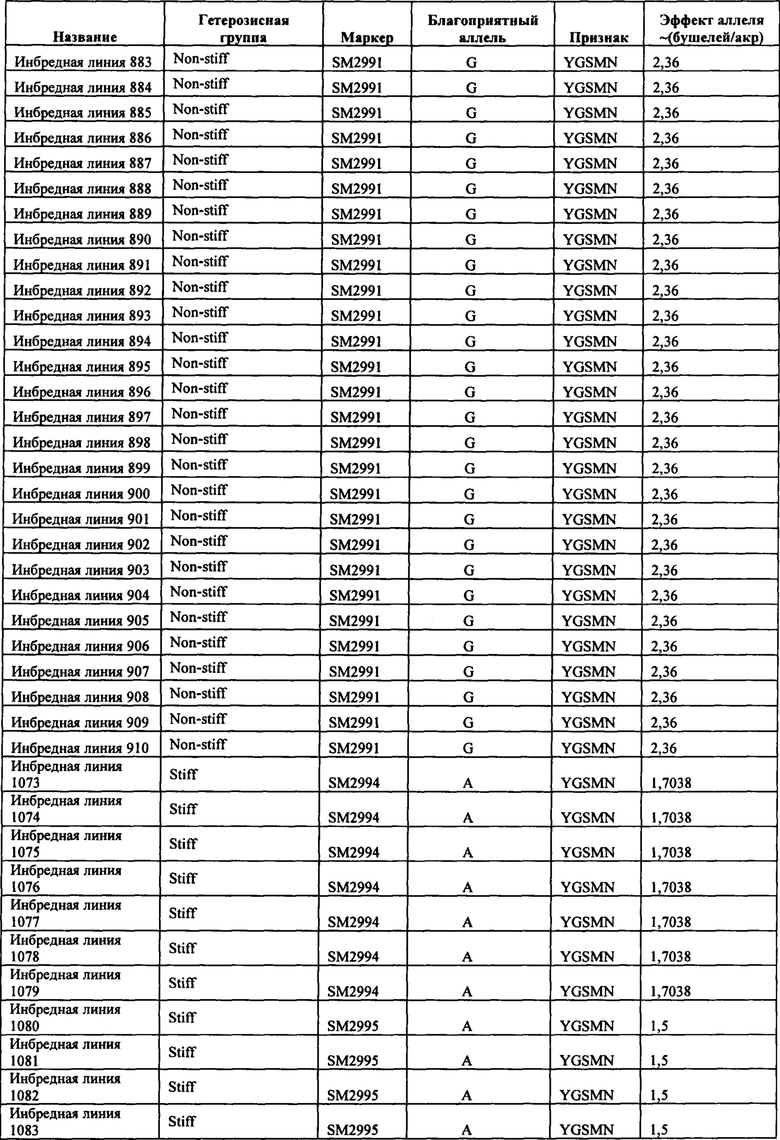
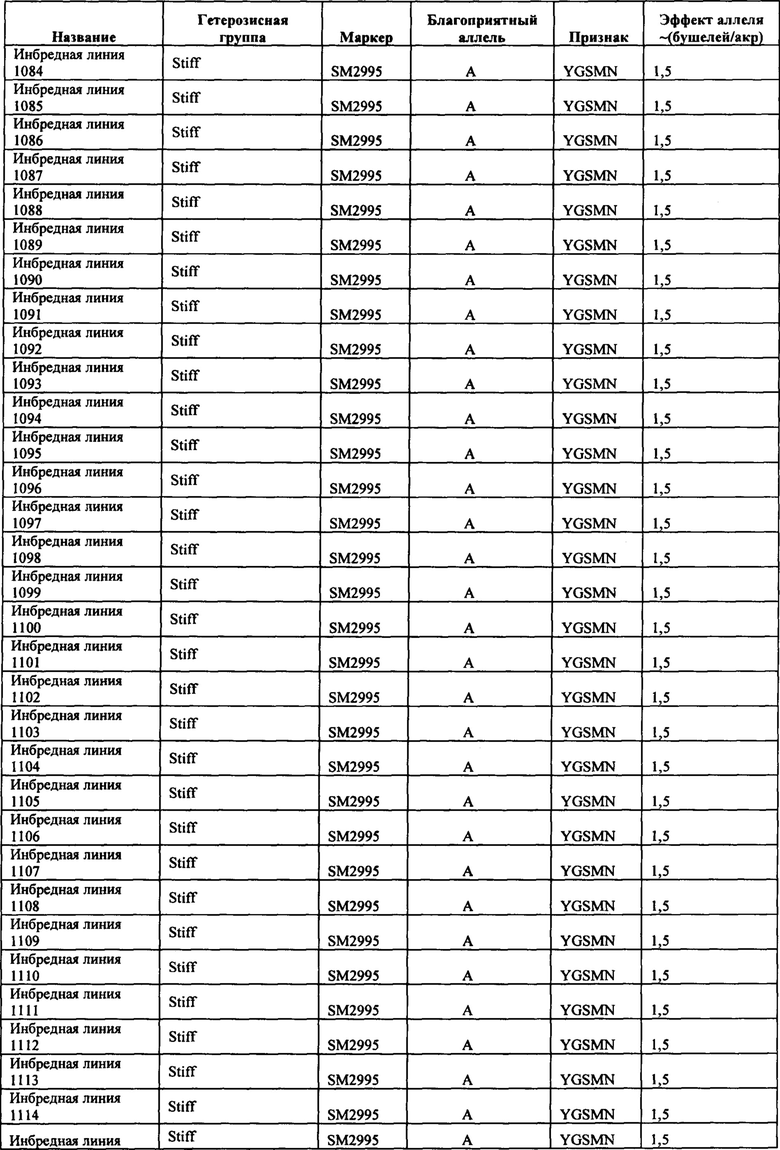
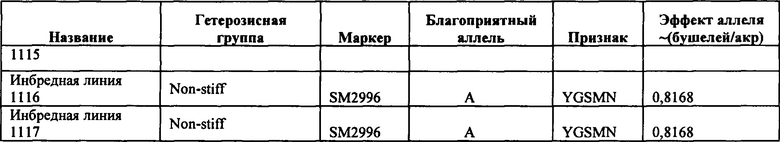
Пример 2. Исследования ассоциации у гибридного маиса
Чтобы оценить воспроизводимость этих результатов в гибридном окружении, генотипические и фенотипические данные для гибридов (урожайность в условиях засухи) применяли для поиска ассоциаций с применением идентифицированных SNP (см. таблицы 12-13).
Две гетерозисные группы, Non-stiff stalk (NSS) и Stiff Stalk (SS), анализировали отдельно. В случае каждой гетерозисной группы два различных набора фенотипических данных анализировали в отношении 1) урожайности в условиях стресса, вызванного засухой, в бушелях/акр, измеряемой в испытаниях с управляемыми условиями стресса (MSE); и 2) урожайности в условиях стресса, вызванного засухой, в бушелях/акр, измеряемой в испытаниях с целевыми условиями стресса (TSE). В ходе испытаний MSE количество воды для растения строго регулируют, чтобы убедиться, что стресс, вызванный засухой, происходит во время цветения, в противоположность испытаниям TSE, при которых растения выращивают в местах произрастания с низким уровнем осадков, и количество воды для растения регулируют частично, что приводит к умеренному стрессу, вызванному засухой, на протяжении всего периода вегетации. Популяции из 24 родительских линий применяли для получения семейств (линий потомства), применяемых в анализах NSS. В общей сложности эти родители дали 167854 варианта, сегрегирующих из них. 24 родительские линии секвенировали с применением подхода сокращенного геномного секвенирования нового поколения. Объединение генотипических и фенотипических данных, полученных в анализе NSS-MSE, привело к тому, что 24 подвергнутые скрещиванию родительские линии дали 45 популяций, в которых в суммарном итоге было 1040 семейств. Затем данные семейства скрещивали с двумя тестерами. Популяции, имеющие менее 10 семейств, исключали из анализа, поскольку они могли обеспечить незначительную дополнительную ценность. Аналогично после объединения генотипических и фенотипических данных для анализа NSS-TSE получали 24 родительских линий, 46 популяций и 1138 семейств. И снова, повторы из данных семейств затем скрещивали с двумя тестерами для получения гибридов, которые подвергали фенотипированию. Двадцать родительских линий применяли для получения популяций и семейств для наборов данных для SS. Среди данных двадцати родителей 112466 вариантов были сегрегирующими. Аналогично наборам данных для NSS родительские линии секвенировали с применением подхода сокращенного геномного секвенирования нового поколения. После объединения этих генотипических данных с фенотипическими данными получали 23 популяции и в суммарном итоге было 553 семейства, для которых были доступны генотипические и фенотипические данные. Повторы из этих семейств затем скрещивали с двумя тестерами для получения гибридов, которые фенотипировали. При объединении генотипических данных с фенотипическими данными получили 23 популяции, и в суммарном итоге было представлено 631 семейство (линия потомства). И снова, особей из каждого семейства скрещивали с двумя тестерами для получения гибридов, подвергаемых фенотипированию.
Тестируемые модели
Две изначальные тестируемые модели представляли собой модель фиксированного эффекта с условием взаимодействия (1), тестируемую с применением PROC GLM в SAS, и модель случайного эффекта с условием взаимодействия (2), тестируемую с применением PROC Mixed REML в SAS.
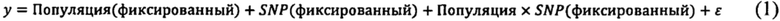
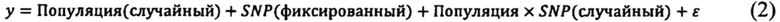
Различие между данными моделями заключается в том, обрабатываются ли популяция и соответствующее условие взаимодействия, как фиксированные или случайные. Если условие популяция определено как фиксированное, тогда отбирают результаты, специфические для популяций. Если условие популяция определено как случайное, тогда считается, что популяции, включенные в анализ, представляют собой случайную выборку из более крупной группы популяций.
Для генотипирования панели ассоциаций на основе семейства применяли MaizeSNP50 BeadChip (Illumina, Сан-Диего, Калифорния). Идентифицировали маркеры, сцепленные с локусами оптимизации потребления воды SM2987, SM2996, SM2982, SM2991, SM2995, SM2973, SM2980 и SM2984, характеризующиеся значимыми ассоциациями с повышенной урожайностью в условиях засухи (маркеры и отрицательный логарифм Р-значения для ассоциации можно найти в таблицах 1-7).
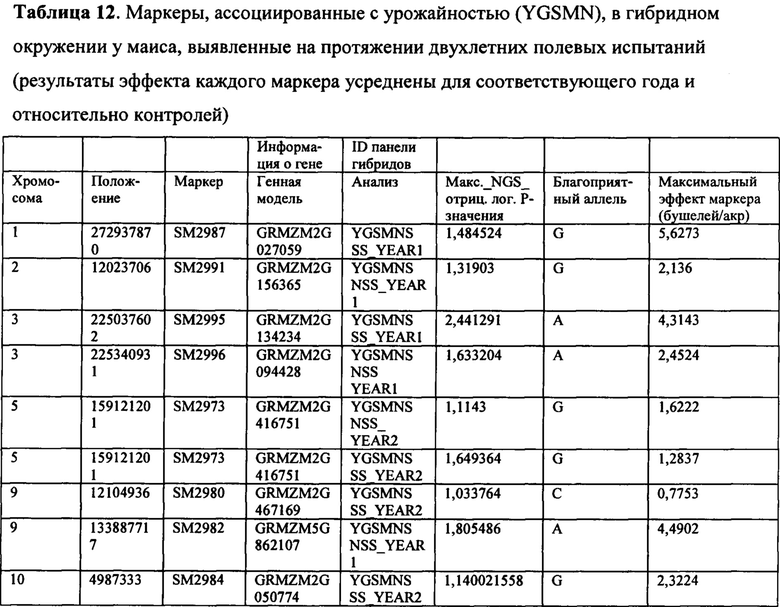
Пример 3. Трансгенная экспрессия генов урожайности маиса
Создавали трансгенные растения Arabidopsis, которые конститутивно экспрессировали следующие гены маиса: GRMZM2G027059 (SEQ ID NO: 1); GRMZM2G156365 (SEQ ID NO: 2); GRMZM2G134234 (SEQ ID NO: 3); GRMZM2G094428 (SEQ ID NO: 4); GRMZM2G416751 (SEQ ID NO: 5); GRMZM2G467169 (SEQ ID NO: 6); GRMZM5G862107 (SEQ ID NO: 7); GRMZM2G050774 (SEQ ID NO: 8). Эксперименты и результаты обобщены ниже.
Методология
Предсказанную кодирующую последовательность для каждого из генов маиса синтезировали и клонировали в бинарный вектор, управляемый 35s промотор без оптимизации кодонов.
Трансформацию Arabidopsis проводили как описано в Zhang et al. (2006), с применением штамма GV3101 Agrobacterium. Затем Agrobacterium, несущих конструкцию, трансформировали в экотип Col-0 Arabidopsis. Семена Т0 подвергали скринингу на среде MS, содержащей 0,6% PAT. РАТ-устойчивых трансформантов Т0 подтверждали с помощью анализа Taqman©, а затем переносили в теплицу для получения семян Т1.
В теплице поддерживали условия фотопериода из 10 часов светового дня в течение первых четырех недель и фотопериода из 16 часов светового дня во время цветения. Поддерживали интенсивность освещения примерно 6000 люкс и температуру около 24°С на протяжении дневного времени и 20°С на протяжении ночного времени. Поддерживали влажность около 40-60%. Растения выращивали в смеси почвы с питательными веществами и вермикулита 1:1.
Экспрессия белков
Для исследования экспрессии белков все представляющие интерес гены сливали с GST на их N-конце и клонировали в вектор экспрессии. Векторами экспрессии трансформировали Е. coli, с применением стандартных процедур трансформации, и клетки выращивали в среде LB до OD600, составляющей 0,8. Экспрессию индуцировали путем добавления IPTG (изопропил-бета-D-1-тиогалактопиранозид) до конечной концентрации 0,4 мМ. Клетки инкубировали при 16°С со встряхиванием в течение 16 часов. Клетки осаждали с помощью центрифугирования и ресуспендировали в 20 мМ Tris-HCL, рН 8,0, 500 мМ NaCl и дополняли полной смесью ингибиторов протеаз (Roche). Клетки лизировали с помощью ультразвуковой обработки, и осветленный лизат связывали партиями с GST-агарозой (GE Life Sciences). Смолу тщательно промывали с помощью 20 мМ Tris-HCL, рН 8,0, 500 мМ NaCl, и связанный белок элюировали в буфере для промывки, содержащем 10 мМ глутатиона (Sigma). Элюированный белок разводили в 20% (объем/объем) глицерине и хранили при -20°С.
Измерение содержания хлорофилла
Образец ткани листа трансгенных объектов Arabidopsis и контроли дикого типа отбирали в количестве 0,01 г с 3 повторами для каждого. Образцы листьев измельчали и добавляли 800 мкл ацетона. Эту смесь помещали в темноту на два часа, а затем осаждали центрифугированием. Затем жидкую часть анализировали на спектрофотометре при 663 нм и 645 нм. Общий хлорофилл (мкг/мл) рассчитывали согласно следующей формуле:
Общий хлорофилл (мкг/мл) = хлорофилл а + хлорофилл b = (20,2 X А645) + (8,02 X А663)
Анализ эстеразной активности
Эстеразную активность анализировали как описано в Brick et al. (1995). Смесь для анализа инкубировали в лунках микротитрационного планшета при комнатной температуре в течение 50 минут. Гидролиз р-нитрофенил-ацетата (pNP-Ac, Sigma, № по кат. N8130) и образование р-нитрофенола отслеживали с помощью спектрофотометра по возрастанию поглощения при 400 нм. Смеси для анализа без Смеси для анализа без субстрата или фермента включали в качестве контролей. Контрольный субстрат (субстрат, инкубируемый без фермента) также применяли из-за спонтанного деацетилирования pNP-Ac.
Анализ метаболитов
Растения выращивали в почве в течение 4 недель при фотопериоде с 10 часами светового дня. Отбирали образцы листьев и проводили измерение общего свежего веса (~1 г). Затем образцы листьев измельчали в порошок пестиком в ступке в условиях жидкого азота. Затем измельченный в порошок материал лиофилизировали с помощью Freeze Dryer EPSILON 2-4 LSC согласно следующей процедуре: основная сушка (-10°С, 0,4 мбар в течение 2 дней) с последующей окончательной сушкой (40°С, 0,1 мбар в течение 6 часов). Порошок переносили в полипропиленовую пробирку для отправки. Анализ метаболитов проводила компания Metabolon, Inc., США.
A. Ген GRMZM2G027059 (SEQ ID NO: 1), предположительно вовлеченный в контроль содержания хлорофилла
Полагают, что GRMZM2G027059 кодирует 4-гидрокси-3-метилбут-2-енилдифосфатредуктазу, которая является важнейшим ферментом для биосинтеза фотопигментов (таких как хлорофиллы и каротиноиды) и гормонов (гиббериллины и ABA). Не ограничиваясь теорией, полагают, что растения, сверхэкспрессирующие или несущие данный ген, могут быть более устойчивыми к абиотическому стрессу (например, засухе) по сравнению с растениями с контрольным геном.
GRMZM2G027059 экспрессировали в Arabidopsis (конструкция 23294), и содержание хлорофилла измеряли, как описано выше. Как показано на ФИГ. 1, содержание хлорофилла в трансгенных растениях было существенно выше, чем в контрольном (СК) растении (см. ФИГ. 1). Это исследование подтверждает, что GRMZM2G027059 действительно играет роль в повышении содержания хлорофилла и это, в свою очередь, может представлять собой возможный способ действий для получения растений с повышенной урожайностью в условиях засухи и условиях, отличных от засухи. Не ограничиваясь теорией, другая возможность заключается в том, что сверхэкспрессия GRMZM2G027059 также может повышать выработку гормонов восприимчивости, например, повышение реакции на стресс с участием ABA.
B. Ген GRMZM2G156365 (SEQ ID NO: 2), предположительно вовлеченный в развитие и поддержание структуры клеточной стенки
GRMZM2G156365, вероятно, функционирует в качестве регулятора структуры за счет модуляции точного состояния ацетилирования пектина (т.е. является вероятной пектинацетилэстеразой). Данное ацетилирование будет воздействовать на ремоделирование и физико-химические свойства клеточной стенки, с воздействием тем самым на способность к растяжению пыльцевой клетки. Не ограничиваясь теорией, возможно, что снижение экспрессии данного гена могло бы повышать прорастание пыльцы в условиях абиотического стресса (например, засухи).
Сверхэкспрессия GRMZM2G156365 изменяет содержание глюкуроната, ксилозы и 3-дезоксиотулозоната у трансгенных растений (см. ФИГ. 2). Все они представляют собой остатки cахаров, вовлеченные в образование пектина. Немного большее содержание глицерина выявляли в трансгенных объектах в отличие от контроля дикого типа, это может быть обусловлено эстеразной активности, за счет которой высвобождается глицерин.
C. Ген GRMZM2G134234 (SEQ ID NO: 3), вовлеченный в регуляцию абиотического стресса
Ген GRMZM2G134234 маиса кодирует предполагаемый транскрипционный фактор семейства DUF1644, исходя из анализа аминокислотных последовательностей. Известно, что данные типы генов усиливают засухоустойчивость и устойчивость к засолению у других сельскохозяйственных культур, таких как рис. Полагают, что GRMZM2G134234 мог положительно регулировать гены, обеспечивающие реакцию на стресс, для повышения стрессоустойчивости маиса во время периодов стресса. Не ограничиваясь теорией, растения, сверхэкспрессирующие GRMZM2G134234, могли быть более устойчивыми к абиотическим стрессам, таким как засуха и засоленность.
D. Ген GRMZM2G094428 (SEQ ID NO: 4), предположительно вовлеченный в биосинтез лигнина и поддержание структуры клеточной стенки
Ген GRMZM2G094428 маиса кодирует предположительную ацилтрансферазу BAHD, исходя из анализа аминокислотной последовательности. Таким образом, данный ген мог быть ответственным за р-кумароилирование монолигнолов в биосинтезе лигнина, этерификацию феруловой кислоты (FA) до глюкуроноарабиноксилана (GAX) в клеточной стенке. Сверхэкспрессия данного гена может повышать содержание лигнина, что может придавать растению устойчивость в условиях абиотических стрессов, включая засуху и засоленность. Не ограничиваясь теорией, снижение экспрессии ацил-соА-трансферазы BAHD может снижать содержание FA или рСА и изменять содержание лигнина.
Результаты указывают, что в трансгенном растении Т1 содержание р-кумаровой кислоты (рСА) и синаповой кислоты (SA) снижается, а спермидина повышается (см. ФИГ. 3). Белок GRMZM2G094428, по всей вероятности, вовлечен в образование клеточной стенки. Сверхэкспрессия данного гена в трансгенном растении изменяла содержание компонентов, связанных с клеточной стенкой.
E. Ген GRMZM2G416751 (SEQ ID NO: 5), предположительно вовлеченный в образование экзины
Потеря урожая, вызванная стерильностью пыльцы, что обусловлено засухой, является главным фактором в товарном сельскохозяйственном производстве. GRMZM2G416751 мог быть вовлечен в образование экзины пыльцы, и при этом растения, сверхэкспрессирующие данный ген, могли избегать стерильность пыльцы в условиях стресса, вызванного засухой.
Результаты указывают на то, что при сверхэкспрессии GRMZM2G416751 показано снижение содержания метаболитов, необходимых для образования клеточной стенки (см. ФИГ. 4). Профили метаболитов указывают на то, что у трансгенных объектов снижено содержание нескольких метаболитов, необходимых для образования клеточной стенки, таких как глюкуронат и 3-дезоксиотулозонат для пектина, р-СА для кутана и лигнина и синапата для биосинтеза лигнина. Дополнительный анализ с мужскими репродуктивными тканями, такими как пыльца или пыльник, необходим для оценки роли генов в образовании экзины пыльцы.
F. Ген GRMZM2G467169 (SEQ ID NO: 6), предположительно вовлеченный в регуляции ретроградной передачи сигнала
В условиях различных биотических и абиотических стрессов сигналы, такие как окислительно-восстановительный дисбаланс в PS1, возникают в хлоропласте и передаются в ядро для контроля паттернов экспрессии генов (ретроградная передача сигнала). GRMZM2G467169 кодирует предполагаемый полиаденилат-связывающий белок, который мог бы регулировать ретроградную передачу сигнала для повышения стрессоустойчивости маиса. Растения, в которых сверхэкспрессируется данный ген, могут быть более устойчивыми к абиотическим стрессам, таким как засуха.
Данные указывают на то, что сверхэкспрессия GRMZM2G467169 повышает содержание хлорофилла по сравнению с контролями (см. ФИГ. 5).
G. Ген GRMZM5G862107 (SEQ ID NO: 7), предположительно вовлеченный в модулирование экспрессии генов, реагирующих на тепло, и/или целевых генов
Ген GRMZM5G862107 маиса кодирует предполагаемый белок S1, связывающий 30S рибосомальную РНК, исходя из анализа аминокислотных последовательностей. GRMZM5G862107 мог быть ответственным за холодовой и тепловой стрессы за счет модулирования генной экспрессии генов, реагирующих на тепло, и/или их целевых генов.
Данные указывают на то, что белок GRMZM5G862107 вовлечен в регуляцию экспрессии HsfA2. HsfA2 имел относительно высокий уровень экспрессии в 23292 по сравнению с контрольными растениями дикого типа (см. ФИГ. 6).
Н. Ген GRMZM2G050774 (SEQ ID NO: 8), предположительно вовлеченный в защитные реакции растения
Ген GRMZM2G050774 маиса кодирует предполагаемую ATL6-подобную Е3-лигазу с доменом RING-палец, исходя из анализа аминокислотных последовательностей. В случае Arabidopsis было обнаружено, что ATL6/ATL31 играет критическую роль в реакции на баланс C/N, а также в защитной реакции растений. Сверхэкспрессия ATL6/ATL31 может позволить растению хорошо расти в условиях недостаточного снабжения N и проявлять повышенную устойчивость к Pst. DC3000. 14-3-3χ (также известный как GRF1) идентифицировали как мишень для ATL31. Не ограничиваясь теорией, возможно, что GRMZM2G050774 может играть роль в эффективности утилизации азота у растений, и сверхэкспрессия указанного гена позволяет растению лучше адаптироваться к условиям сильного стресса (например, такого как засуха или тепловой стресс).
Будет понятно, что различные подробности объектов, раскрытых в настоящем изобретении, могут быть изменены без отклонения от объема объектов, раскрытых в настоящем изобретении. Кроме того, вышеизложенное описание приведено только с целью иллюстрации, а не с целью ограничения.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ЗИНГЕНТА ПАРТИСИПЕЙШНС АГ
<120> УЧАСТКИ ГЕНОВ И ГЕНЫ, АССОЦИИРОВАННЫЕ С ПОВЫШЕННОЙ УРОЖАЙНОСТЬЮ
У РАСТЕНИЙ
<130> 80995-US-L-ORG-NAT-1
<160> 77
<170> PatentIn версия 3.5
<210> 1
<211> 2115
<212> ДНК
<213> Zea mays
<400> 1
caagaggaca gcaaccggcg gccctcgcag ccgcgcctca cgcgccacgg aatatccctc 60
cagttccacg gggggccacg gcgtcagaac tcagaaggcc gcagggataa gagagagcgg 120
gtcccgtccg agccgaggca gcccattcgc cgtccgcccc gcctcttcct gccgccgagc 180
gccacgaggc ccacgcccgc gatggcgact atcacgacgc cgctccgctc cgctctgttc 240
tctccggccg cctcgtccgc gggccgccac cgcgggggcc ggcgccgcgc gccctcctcc 300
gtgcgctgcg acgcctcccc gccctcgcac gccgcggccg cctcgctcga cccggacttc 360
gacaagaagg cgttccgcca caacctcacg cgcagcgaca actacaaccg caaggggttc 420
gggcacaaga aggagacgct cgagctcatg agccaggagt acaccagcaa cgtcatcaag 480
acgctcaagg agaacggcaa ccagtacacc tggggccccg tcaccgtgaa gctcgcggag 540
gcctacgggt tctgctgggg cgtcgagcgc gccgtgcaga tcgcgtacga ggcgcgcaag 600
cagttccccg aggagcgcat ctggctcacc aacgaaatca tccacaaccc caccgtcaac 660
aagaggttgg atgagatggg tgtagaaatc attcctgttg acgcgggtat caaggatttc 720
aatgtcgtcg agcaaggtga tgttgttgtg ttgcctgcat ttggagctgc tgtggaggaa 780
atgtacacgc taaatgagaa gaaggtgcag attgttgata cgacatgccc ttgggtttca 840
aaggtctgga atatggtcga aaaacacaag aagagtgaat atacttcaat tattcatgga 900
aagtattccc atgaagaaac tgttgccact gcttcttttg caggaaagta catcattgtg 960
aagaatatgg cagaggcaac ctatgtgtgt gattatatac ttggtggcca acttgatggg 1020
tctagctcaa caaaagagga gttccttgag aaattcaaga aagctgtttc tccagggttt 1080
gatcctgatg ttcatcttga tatggtggga attgcaaatc aaacaacaat gcttaaagga 1140
gaaactgaag aaattgggaa gcttattgaa aagacgatga tgcaaaaata tggagttgaa 1200
aatgtaaacg atcacttcat ggccttcaat actatttgtg atgccactca ggaaagacaa 1260
gatgctatgt atcagctggt gaaagagaaa gttgacctta ttcttgttgt tggaggatgg 1320
aattcaagta acacctctca tctgcaagaa atcggagaac tcagtggaat tccatcatac 1380
tggattgaca gtgaacaaag gattggacca ggaaacagga tcagctacaa gttaaatcat 1440
ggtgaactgg ttgagaaaaa taactggtta cccgaggggc ctattaccat tggtgttact 1500
tcaggtgcct caactccaga taaggttgtt gaggatgctc ttcagaaggt atttgagatc 1560
aagcgtcagg aaattttgca ggttgcataa attttaagca gagatttggt gaagagctga 1620
atagttttgg cttggcaaag gttactagaa acgttgcaca ggcaaatgtt tgtacagtag 1680
ctaaggatgt aacgagttgg gcacgaatac taccacgagt cactatcctt gtgctggaat 1740
ttacagtacg gtggaaacta aaatggtgtt atcattggcc cgaataacat ttgcatcagc 1800
ctttctaaag tctaactttt tgccacttaa attgatgtca gggaagacac tcagatgtgt 1860
aagttaaaga atgcacagtt cctatgtgta aaaagcttag ttccgaggag gagatggcgc 1920
cccttccttt cccgcctgac cgttcctatc ccctgaccct ctcctctacc cccgacgcct 1980
cttcaccttc ctccaccaac cccacgccgg agaccactcc gatcccggcg gccaatcacc 2040
tctccttccc cggcgtcggc ggggccctct gtttgtggtc ggtccaaggc gcagcggtgg 2100
tgtgacgaca gcgca 2115
<210> 2
<211> 1961
<212> ДНК
<213> Zea mays
<400> 2
gcatgaccac tgaattgctc gagtgcatat atgatcggat cctccagtga tctgattgat 60
gctcaaagaa tccatgcact atgcaggtag gtggatcgat agctgggagg cattaaagcg 120
gacgatgacg ccttggcctc gctgcaatct tgcagattgc tgctgcagcg cttctttaag 180
agccaaccat ccacatatac cttgcttgac gccaagccac cggcattcca ctcccagcag 240
cggtgggaac aaagagtgcc agcgtctcac ccctgaggcc gcgcagagcc actgcttgcg 300
tctctgtctc tctgtctgct cgtttttctc cttctgcgac tcgtaggagg gtgaggtctt 360
gcctcgcgga atggcggcgt ccggggcatg gctggcccgt gcgacggcga cggcggtgct 420
gggtttcgtc ctggcggtgg cgtcagctga ggcggcatcg ggggacgtgg agatggtgtt 480
cctcaaggcc gcggtggcca aaggcgcagt gtgcttggac ggcagcccac cggtgtacca 540
tttctctccc ggctccggtt ctggcgccaa taactgggtc gtccacatgg agggaggagg 600
gtggtgcagg aatcctgatg agtgtgctgt ccgcaagggc aacttcaggg gctcctccaa 660
atttatgaag ccactctcgt tttcagggat attaggcggc aaccaaaaat ccaatcctga 720
tttctacaac tggaatagag taaagatcag atactgtgat ggttcatcat ttactggtga 780
cgttgaggct gtggacactg cgaaagatct ccgttacaga gggttcagag tctggcgtgc 840
cgtcatcgat gatctactta ctgtgagggg aatgagcaag gcgcaaaatg ctcttctttc 900
tggatgctca gccggaggtc tagcagcaat actacactgt gacagattcc atgatctgtt 960
tccagcgaaa acaaaggtca agtgtttttc tgatgctgga tattttttcg atgggaagga 1020
tatctccggg aacttttacg ctaggtcaat ctataagagc gttgtgaatc tacatggatc 1080
agccaaaaat ttaccagctt catgtacctc aaagccaaag caatcacctg agctgtgtat 1140
gttcccacag tatgttgtcc cgacaatgcg cacaccattg ttcatactta atgcagccta 1200
cgattcgtgg caggtcaaga acgtcctagc acctagtcca gctgatccga agaagacttg 1260
ggcccaatgc aagcttgaca tcaagagctg ctccgccagc caactcacaa ccttgcaaaa 1320
tttcaggaca gattttctgg cagcactccc taaaacgcag tctgtaggca tgttcatcga 1380
ctcatgcaat gctcactgcc aatcaggatc tcaagacacg tggctagccg atggttctcc 1440
cacggttaac aagacgcaaa ttggcaaggc ggtgggggac tggtactacg atagggaggt 1500
ccctcggcag attgattgcc cgtatccctg caacccaact tgcaagaacc gtgatgatga 1560
ttgagcaatt gtataagtag ttcatgttat cgaaatgaaa acaataaagg atcacaacgc 1620
gcgcccgtag ttgtagatga tgaattataa acacatatga ctgagctcaa agttgtttaa 1680
tcatcatctg ttgcgaaatg aggaagacaa ttggtgtctt gaagctgtgt tttcgactgt 1740
gtctaaagcg taaatgtaac gtatattgtg tcttcgccta tgcttaagac attggactag 1800
ttgattggtc aatttaattt attaaatgtt ttgattggtg taatgaatat aataagtcgt 1860
gcatgccgca tgactaggct tccagtcttc cacttacacc ggctaagcac tgtctatata 1920
tatgtagtca ctttggatca atgaatcagc tgtttttatc a 1961
<210> 3
<211> 2149
<212> ДНК
<213> Zea mays
<400> 3
gagcgagcca ccacccaacc tgaccccttc gccccgtatc gttcccttcc tcctctcttt 60
tccccaccgc cccctttctt ggcgtctacc cggcgcgacc aggaccgaat cctaaggccg 120
ccggccgccg ccgccgccgc ctgctcccgt tggacacggt aaaacctcct ccctcctcgt 180
ccgtgtcgcc ggggtccggg gtccaagcgg ctgcgcgcgc ggtctcgccg ccggcgccga 240
tctgggcgcc gccggcgttg accctgtccg attcgccccg ggctgcgaga cctctgcctc 300
cctgaccggt tactcggaac ttctactcgc ctgtgggatc ctccagcgga tcagatgagc 360
acatcgactt gagcaccacg ctttttggtt ggaaggcgaa tcgtagagct ttcctgggtt 420
tctctgaggg ctcttctcag atgtggcgtc ctccagagtc taccacatac tgtttgagga 480
gtcctggttc ttttcggaat ccggactaac caagggctcc tactgtgcac gactgcttga 540
caggatattt cggattatta ttatgtgcgt gcgagcgcgc gtgtgcgcct tctagaggca 600
tgattcttaa atcagagcct tcgtgtttaa atccgagttt gccttcgtgt tgaactatga 660
gtgaatttca ttttctgcgg gagttgaagt cgatttagat caggacagtg tttcttgcga 720
tctgattaag cctttttttt tcttgctatt gtgatttctt ttttcagagt ttggagtaaa 780
gaaaccaacc ctgcatctgt attctgtctg tctgtgctgc ttcgaataag ccttgcatct 840
cgctgacttg ggatataact atgccgaagg acaggagctc ccgcgtttcc tcttatgaga 900
gccgccgggc tggtgcctcc ccatacttct catcgtctca tggacagagc agttcttgtc 960
gccggtccga ggagtcttgt ggggcagcag cggcggcagc agcaaagcaa gctgcagagt 1020
gggaggatgt tcggtgcccg gtgtgcatgg accacccgca caacgccgtc ctgctggtct 1080
gctcctcaca cgagaagggc tgccgcccct tcatgtgcga caccagctcg cggcactcga 1140
actgctatga ccagtaccgg aaggcctcca aggattcaag gacagagtgc agcgagtgcc 1200
agcagcaggt tcagctctcg tgcccactgt gccgtgggcc ggtcagcgat tgcatcaagg 1260
actacagcgc gcggaggttc atgaacacca aggtccggtc gtgcaccacg gagtcgtgcg 1320
agttcagggg cgcctaccag gagctgagga agcatgctag ggtggagcat ccaacaggaa 1380
ggccaatgga ggtagaccct gagcggcagc gggactggcg ccggatggag cagcaacggg 1440
accttggaga cttgatgagc atgctgcgtt cagggttcaa cagcaatatt gaggacgaca 1500
gtggcgggct tggagacacc gaagaagggg gagaggaagc tgaaatgact ccggcctcca 1560
taaccatggt cttcatcatg ccatctagag gctcaatcat gcagtaccta tcggaacgca 1620
gcagaacgat cattctggtc agtcggaggc gagcaagcag cagcagcggt ggcgacgctg 1680
aagccactgc tccagacagc gaggaaggtg atgaccctat gccatcggca gaggcatctg 1740
ctggttcaca gcattcttcc gaacaagagg aggctgacgg tgaccctgcc caatgacgta 1800
agtcagctgg caagaggtgt gccatggcat cttcttagcc tgaagatccc gaccaacatg 1860
gcaacatggt atgtggcgaa gaaaaacatg gatgcaggtg ccaccaaggc gtagccagga 1920
caatctgtct acgcaggaaa atcgagggga atcagcatcg cgcgaaggct tcagaaacgt 1980
ggtggcctct agtaccaatt ctaatgtttc cgcggggttc tgtggcaatg gagagagaga 2040
aacaattggg tggtagctat cttgtctgaa tggatttcat tttccttgca ttgtaattct 2100
ctaatatata ttatcatatg aaatagattc ggccgtattt gcactgcgt 2149
<210> 4
<211> 1747
<212> ДНК
<213> Zea mays
<400> 4
gtcagctaag cgaacgtctc gattcgtcaa agtctgaaat taagctggac accgctcccg 60
caccaaacca aactaattac ctcttcccgc gacctcctcc cccggcgtac ctccggagat 120
ccaccccgac ccaccaccgc gtgcctcggc aatggccgcc gctccgacca ccgtaaccaa 180
gtccccgccg tccctggtcc cgccggcggg gcccacgcca ggcggttccc tcccgctctc 240
ctccatcgac aagactgccg ccgtccgcgt ctccgtcgac ttcatccagg tcttccccgc 300
ccctacgtcg gggaaggagg accggagccc ctcctccacg atcgcggcta tgcgcgaggg 360
ctttgccaag gcgctcgtgc cgtactaccc cgtcgccggc cgcatcgccg agcccgttcc 420
gggggagcct gagattgagt gcacagggga aggggtgtgg ttcgtggagg ccgaggccag 480
ctgctccctc gaggaggcgc ggaacctcga gcgcccgctg tgcatcccca aggaggagct 540
gcttcctcgt ccgccggccg gggtgcgcgt ggaggacacc ctgctgctcg ctcaggttac 600
aaagttcaca tgtggtggat ttgctgtggg catttgcttc agtcacttgg tgttcgatgg 660
gcagggtgct gcacaatttc tgaaagcggt gggtgagatg gctaggggcc tccctgagcc 720
atcgatcaag ccaatctggg ctcgtgatgc catccccaac ccacctaagc cacccctagg 780
tccgccgccg tcattcaccg cattcaactt tgagaaatcg gttcttgaga tctctccgga 840
cagcatcaag aacgtgaagg atcaggttgc aagtgaaacc aaccagaagt gttccacttt 900
cgacgtggtc actgccataa tcttcaaatg ccgcgccttg gcagtcgact tcgcgcccga 960
cgctgaggtc cgcttgggct tcgcagccag cactcgccac ctgctgagca atgtgctgcc 1020
ctcggtcgaa ggctactacg ggaactgtgt gtacccaggt ggtctcacca agaccagcca 1080
ggaggtgaag gaagcttcgc ttgtggagat cgtgaccgtg atcagggaag ccaaggaagc 1140
tctgtcatcg aggttccttg actggttgag cggcggcgcc aaggagaacc actacaacgt 1200
gtcgctagac tatggcaccc tcgtcgtgac tgactggagc catgtgggct tcaacgaggt 1260
ggactacggg ttcggtgagc cgagctacgt gttcaccctg aacgacgacg tgaacatcgt 1320
cccctccgtt gtgtacctga agccgcccaa gccgaagcag ggcatcaggc tggtcctgca 1380
gtgcgtggaa ggccatcact ctgccgtgtt cggcgaggag ttgcagaagc atgcatagag 1440
tgagtgtatt ctacagtggg aatctgttgt attttatttg ttgtgtcaaa ttgctgctcc 1500
cggaatttgc ttgcaataag gcagattggt cgtgtttata ctttgtacca ttatcagcac 1560
gttacattat acatgtgatg aatattgaca gtgacgaaag aataataatg ttcccatttg 1620
gaacaaatta tttcagattc gttggcctgc tgtaggttcc tggtgtctcg agttttaacg 1680
tgtgtaactg tgttatgtat aagtataact ctgacagtgt ttgatgattg atcaacggca 1740
gaaagaa 1747
<210> 5
<211> 2311
<212> ДНК
<213> Zea mays
<400> 5
tctcattcaa gtgctgtaaa catataaccc aaatatgatc atttttttgt gttctcatta 60
tttcttttct aagaagtaag acccaaccag ggttttttgt ccatcattaa gtgattgttg 120
ctgattgaat acagaagtta cagaagaaga agctgaagaa aaattgcaag acacaataag 180
ggagaggttt tcgtcctttg gtgaggatta ccatgctgtt gatattctat tagcagagat 240
gatgtgtatg aactttttgc tttcaagcat tgtgtgggaa gaaggataca gcttgccctt 300
tgtaaagaac ttgatgagag gatgcatgac ctgaaaaagg aactggaggg ttacaatact 360
ggagattttg atgaaactaa caagaagaaa gctcttgatg cactgaagag aatggaaagc 420
tggaacttat tcagagatac ttcagtggaa catcatagtt acacagtggc tcatgattca 480
tttcttgcac aacttggatc tatgttatgg ggctctatga ggcatgtaat tgctccttct 540
gcctctcata gagtgtacca ttactatgag aagttatcgt ttcagttgta ttttgtgaca 600
cgagagaaag tcaggagtat aaagcagtta cctgttaatg taaaatccat cagggagagc 660
ctgaattctg tgctattaca tcatcaaaac tccatgttta gccaaaacat gctgtcattg 720
tcagaggatc catcattgat gatggcattt tcaatggcac gtcgtgcagc tgcggtgccg 780
cttctattag tcaatggcac ctataagtca actgttagca cataccttga ttctgctatt 840
ctccaacatc agctacagaa gctaaatgag cacaattcac tgaaaggaag gcattcaaat 900
cacaggtcaa cattagaggt cccaatattc tggttcatac ataatgaacc catattattg 960
gacaaacatt atcaagccaa ggctctctca aatatggtcg tagtagttca gtcagatgat 1020
gattcctggg aaagccattt gcagtgcaat ggaagaccca tcttatggga tttgaggaaa 1080
ccggttaaag ctgctattgc tgcaactgct gagtatgtat ctggtctact tcctccacat 1140
ctggtttata gccatgctca tgaaactgca attgaggact ggacctggtc tgtgggttgt 1200
aatccctcag ctgtgacttc tgaaggttca caactttcag agttccagca agatgtgatt 1260
gctcgtaact atattattac ttcagtggag gaatccattc aagtaatcaa ttcagcaatt 1320
cagcaattgg taatagagcg gactactgaa aaaggcttca aaattttcaa ggctcacgaa 1380
agtaagatgg ttgagaagta caatgccgtt gttagcttgt ggagaagagt atcggctatg 1440
tccaagggat tgcgatatgg tgatgcagta aaacttatgt caatgcttga ggatgcttca 1500
aatgggtttt ctagtgctgt gaactccacc atttcaagtc tgcaccctgt ccaatgcacc 1560
cgcgaaagga aggtcgacgt gcagctagac ttgacaacac ttcctgcttt tctagctgta 1620
tttttgttgc tttggtttct tctacgtcca aggagaccga agcctaagat caactgaaca 1680
ccgagccaat gagcagcata ggccatagag tttttgtgaa tacgcgcatg gattacagat 1740
ggcgctggag catggcccgg gaattccaaa ggtccaaaac accgggtggc agggaacaag 1800
gtttcagaag attgcaatcc tgacacatcc ccaagttgta gcagagttgg aatgtcatga 1860
aaactttaat tcattcagtc ctgtcctcgt tccgggttta gccaattctt cctcgttccg 1920
ggtaaggcct tgttcgtttg tgtcggattg gtgggtcgga acaattcccg gccggattgc 1980
ttctctaatt tatataaact ttgattagcc ggaacgattc cgggtgcaat ccgacgcaaa 2040
cgaacaagcc ctaactgaga ttaatttgtc cttgctgtaa tgtttagcca gtcctgcccc 2100
gatccgggga actgagagat tgtctttatc gcaacattaa cggctagcgg ttagtatcat 2160
cttccagtca cctggaatgt tactagtaca atccaattgt ctgtttcctg ccgcttacat 2220
gtaaaagtcc atactcaagt tttacagaaa gaaacatgtt ctgtcattta ttacaaaata 2280
aagccaaata gtaaaatgtt atgtgtacgt a 2311
<210> 6
<211> 2397
<212> ДНК
<213> Zea mays
<400> 6
ggagtttctg gtaacgatgc tactatgggt ccaaagcaca cgcttccacc tggtagtgtt 60
acctcttcag ctgaattggc ttctagcgtt ctgaaaggga gcgaggattg ggatgctgat 120
gtaatggata agtattctat tggaaaagaa ggcaaatcta aaaatattga tccagttagg 180
aaggatgatt cagtagcaat cttagaacag ttctttggca atgttttatc gaaaagcggc 240
agcaacctac caacttatgt tgagaaccag ccattgaaaa ctgatgatga catgatcact 300
tctgtgccag aatcatccaa atttgctcat tggtttcttg atgaagactt gaaacctgca 360
gaagacttat cttcaaagag cctgctctcc atgattgtca aaaatgaaaa tccaggtcta 420
gaaaatttaa accatactcc tttatctgat gctgctgccc agaatttatc cccaagagca 480
cctattgata aacttgattc tgcatcagag cttatctcat ttacatcctc tacgcctgcc 540
aatggagttc ttgaacaatg catccattct gatgttccag aggcagttcc tattatgaca 600
tgtgaggatc ttgagcagac gatgttagca caggttagca atagcagctc aactcagata 660
aatgctacaa aggagcaact gactgttatg gatgaaccag ttgccatgca gaaagtaact 720
gtagataatc atgcatcaca acatcttctt tcattgttgc aaaaaggaac agataataag 780
ggagcacctt ccctgggttt ccagagagaa tcaactgatg aacctctgag tgttgacaca 840
aatttaatgg caaatggtgg aatatctgga agtgatccgg ttaacagtgt tgaaaatgtt 900
cctacttctg ggaaggactt gacattggaa gcgttattcg gggctgcatt tatgaatgag 960
ctccactcga aagatgcacc agtttctatt cgaggagcca caactggtgg tcctactgag 1020
tttgcagaga tgggtaaaac tctgttgtca tctagccatg aaggatacta ccctgttgaa 1080
cagaccgtac acttcaacaa tactaaagat gctgctgtcc gtagagaacc aggtattgag 1140
cattcagcag tacctggtct aagtcagggg agtgctagtt ttgacaagaa aggaatggaa 1200
attcatctgc ctgaagaaga taatttgttt accatgagtg attctctgct tggtcaaaat 1260
tctgatattt tggcatcagt aggatccagc agggttgaag ggctattgcc tgaaaaggca 1320
cttgataacc tcagctatag gtttcaaagt cttgtgcctg gtgatgcaga acacattcaa 1380
gtatatggtc ctgatgcact tggatctcat cctcgtgatt ctcagaatat gtatcatctt 1440
ctacagggta ggcctcctat gatagcacct caccctatga tggatcacat tgttaatagg 1500
aaacagccag ctccatttga tatggcacag tcgatacacc atgattctca ccgttctttc 1560
ccatctaatg tgaatcatat gcaacataat cttcatgggc caggggtccc tcacttggac 1620
cctgctggac atattatgcg acaacacatg tccatgcctg gaagatttcc tccagaaggc 1680
ttgccaagag gtgtccctcc atctcagcct gtgcatcaca tggctggtta tagacctgaa 1740
atgggtaatg taaataattt ccatatgcac cctcgccagc ccaactatgg agaatttgga 1800
ttgatgatgc caggtccaga ggtgaggggc aatcatccag aggcgttcga aaggttgatc 1860
cagatggaga tgtcagccag atcgaagcaa cagcaggtgc accaccctgc aatggccgct 1920
ggccgtgtgc ctagtgggat gtacgggcac gagcttgatg cgaaattgag atacagatga 1980
tggatgcctg gatgcttcac tccgtacaga ggacctggag gtgtggtttg ttgtatgtgc 2040
gtggtcactc tttgcccaga ctgctgtgta ttatttctgc taacatggtt tagcatcagc 2100
cgtcggtcgc gactgattgg aggcctgcct cacttgtagg gttgtagcat gtacatctga 2160
acgggtgatg gaacggagtg ggtctaagat ctgtaggagc ggaagtctac cgggaaaagg 2220
ggttatggtg tgctgaatgg aagacgtggc gtcgacgtct tagcagccac atgtgtaatg 2280
acgttttctg tctactgttt ctgacgacta tgcagtttcc attttgtata agctctgtta 2340
tgcaaaagga aaaaaaaaga agaaaaaaaa ctgagtcaga ttaacagatt ggcgaca 2397
<210> 7
<211> 1470
<212> ДНК
<213> Zea mays
<400> 7
ctatcctaaa ccccaccgac cggataacag gacactggca ctgccattcc cgtttcctcg 60
ctcgaacaca caccgaagag agagacagag cgagagagga tggcgtccct ggcgcagcac 120
gtcgcgggcc taccgtgccc ccctctatcc ggcgcgtcgc gccgtcgccc cgcggcgcag 180
aggcggcctc cgtcggcgct tgtgtgcggt acctatgcgc tgaccaagga cgagcgggag 240
cgggagcgga tgcgccaggt gttcgacgac gcctccgagc gctgccgcac cgcgcccatg 300
gagggcgtcg ccttctcccc cgacgacctc gacaccgccg tcgagtccac cgacatagac 360
acggagatcg gctcgctcat taaaggaaca gtatttatga ctacctcaaa tggtgcatat 420
atcgacatcc aatccaagtc tactgctttt ttgcccttag atgaggcatg tcttcttgat 480
atcgataatg ttgaagaggc tggcattcgt cctgggttag tagaagaatt catgataatt 540
gatgagaacc caggtgatga aactttgatt ctaagtttgc aagcaattca gcaagaactt 600
gcatgggaaa ggtgccggca acttcaggcc gaagatgtcg ttgtcacggg taaagtaatt 660
ggtggaaaca aaggaggtgt agtagctctt gtggatgggc ttaagggttt cgttccattt 720
tcgcaagtgt catcgaaaac aaccgccgaa gagctgcttg agaaagaatt gcctctgaag 780
tttgtagagg tcgatgagga acaaggcagg cttgtcctca gtaatcgcaa ggcaatggca 840
gatagtcagg cccagctagg tattggatca gttgtcttgg gaactgtaga gagcctaaaa 900
ccttatggcg ccttcattga catcggtgga atcaacggcc ttctccatgt gagccagatt 960
agtcatgacc gtgttgcaga tatctcaaca gttctgcaac caggagatac cctcaaggtt 1020
atgatactga gccatgaccg tgaaagaggc cgagtcagcc tttctactaa gaagcttgag 1080
ccaacacctg gtgacatgat ccgcaatccc aagcttgtgt ttgagaaggc tgatgagatg 1140
gctcagatat tcaggcagag aatagctcag gcagaggcta tggctcgtgc tgacatgttg 1200
agattccagc cagagagtgg attgaccctc agttcagagg gcatcttagg accattgtcg 1260
tcggatgcac cttcggagga ttctgaagat cgcacagatg aatagaggca gttgacgaag 1320
tgcaccgggt ttgagatatg ggatggcagt tcgtcaagct cattttcaat cgggtggggg 1380
aggcatgacg agatcatttt ctgttcagat cgtgaggtcc gttccagtta ttatccattt 1440
ggattaggaa atagaaaaag taacagggtt 1470
<210> 8
<211> 1285
<212> ДНК
<213> Zea mays
<400> 8
caccagtcca ccgcgccaca ggctccaccc tcccctctcg gcccggctcg atggggtccg 60
gcgtgtcgtc gagcatggcg ctggcgctgg cgggcttctg cttcagcgtc ctcttcatcg 120
tcttcgtctg cacgcgcctc gcctgcgcgc tcgtccgccg gcgccggcgc caggcccgcg 180
cccgcctcgc ggccgccccg ccgctcccgc actacgccca cggctacgcc gaccccgacc 240
ctttcccgtc gttccgcgcc gcccgccacc accaccacgc cccgggcctc gatcccgccg 300
ccttccccac ccgcgcctac gccgccgcac aagcctccga ctccgacgac ggctcccagt 360
gcgtcatctg tctggcggaa tacgaagagg gagacgagct ccgcgtgctg cctccctgca 420
gccacacctt ccacacgggc tgtatcagcc tgtggctggc gcagaactcg acgtgcccgg 480
tctgtagagt ctcgctgctc gtgcctgata ctagtactac ccctgaaagc gaacactctg 540
caccccatcc tcctcctcct cctcatcatc atcatcatct gtccagcata gtgataataa 600
gcccaccaag ctcccccgaa ccgtcgagat cggacccgtg ccgatgcctg ttcgccagcg 660
gtggtgggca ctcgtcaagg gcggcagagg ctcctcctcc tcctcctcct cccagacacg 720
agcccgacca ggtcgtatct ggtccaccac cggcagcaga tggggctagc ggctacagct 780
cgccgttgcc tgaagttatt caccccgctc ctgctcctga aaccaacggg cagacagtac 840
ggaagcaggc gggcagcaga tctactaccc cgctaggccc ctgcaaatag cggccgctca 900
ctctgtgtgg gtgggtgggg tgaacaggtg gtgcgtggta aaagcgaagt agagagaaac 960
aagcgacttg aagaggcctg ggttcgttcg tgtacatacg atcgagaaat cgtttcaggt 1020
cattcattca tccattcatt catccatggg cacatactgt ggtattacgg agtattacgg 1080
tgtacgtgta gtgtgccgca ggagagacga cgcgacggca gcagtgcgtt ttccatatgc 1140
gacgggacgg gagcattcga ggagatgatg gcaatggcat ggttttgtgt actgtacggt 1200
aacatttgtc gctgggaatt aataataaaa aacccgtggc tggctgatgc agcagcagct 1260
gtcttattat ccagccacgc atgtg 1285
<210> 9
<211> 462
<212> БЕЛОК
<213> Zea mays
<400> 9
Met Ala Thr Ile Thr Thr Pro Leu Arg Ser Ala Leu Phe Ser Pro Ala
1 5 10 15
Ala Ser Ser Ala Gly Arg His Arg Gly Gly Arg Arg Arg Ala Pro Ser
20 25 30
Ser Val Arg Cys Asp Ala Ser Pro Pro Ser His Ala Ala Ala Ala Ser
35 40 45
Leu Asp Pro Asp Phe Asp Lys Lys Ala Phe Arg His Asn Leu Thr Arg
50 55 60
Ser Asp Asn Tyr Asn Arg Lys Gly Phe Gly His Lys Lys Glu Thr Leu
65 70 75 80
Glu Leu Met Ser Gln Glu Tyr Thr Ser Asn Val Ile Lys Thr Leu Lys
85 90 95
Glu Asn Gly Asn Gln Tyr Thr Trp Gly Pro Val Thr Val Lys Leu Ala
100 105 110
Glu Ala Tyr Gly Phe Cys Trp Gly Val Glu Arg Ala Val Gln Ile Ala
115 120 125
Tyr Glu Ala Arg Lys Gln Phe Pro Glu Glu Arg Ile Trp Leu Thr Asn
130 135 140
Glu Ile Ile His Asn Pro Thr Val Asn Lys Arg Leu Asp Glu Met Gly
145 150 155 160
Val Glu Ile Ile Pro Val Asp Ala Gly Ile Lys Asp Phe Asn Val Val
165 170 175
Glu Gln Gly Asp Val Val Val Leu Pro Ala Phe Gly Ala Ala Val Glu
180 185 190
Glu Met Tyr Thr Leu Asn Glu Lys Lys Val Gln Ile Val Asp Thr Thr
195 200 205
Cys Pro Trp Val Ser Lys Val Trp Asn Met Val Glu Lys His Lys Lys
210 215 220
Ser Glu Tyr Thr Ser Ile Ile His Gly Lys Tyr Ser His Glu Glu Thr
225 230 235 240
Val Ala Thr Ala Ser Phe Ala Gly Lys Tyr Ile Ile Val Lys Asn Met
245 250 255
Ala Glu Ala Thr Tyr Val Cys Asp Tyr Ile Leu Gly Gly Gln Leu Asp
260 265 270
Gly Ser Ser Ser Thr Lys Glu Glu Phe Leu Glu Lys Phe Lys Lys Ala
275 280 285
Val Ser Pro Gly Phe Asp Pro Asp Val His Leu Asp Met Val Gly Ile
290 295 300
Ala Asn Gln Thr Thr Met Leu Lys Gly Glu Thr Glu Glu Ile Gly Lys
305 310 315 320
Leu Ile Glu Lys Thr Met Met Gln Lys Tyr Gly Val Glu Asn Val Asn
325 330 335
Asp His Phe Met Ala Phe Asn Thr Ile Cys Asp Ala Thr Gln Glu Arg
340 345 350
Gln Asp Ala Met Tyr Gln Leu Val Lys Glu Lys Val Asp Leu Ile Leu
355 360 365
Val Val Gly Gly Trp Asn Ser Ser Asn Thr Ser His Leu Gln Glu Ile
370 375 380
Gly Glu Leu Ser Gly Ile Pro Ser Tyr Trp Ile Asp Ser Glu Gln Arg
385 390 395 400
Ile Gly Pro Gly Asn Arg Ile Ser Tyr Lys Leu Asn His Gly Glu Leu
405 410 415
Val Glu Lys Asn Asn Trp Leu Pro Glu Gly Pro Ile Thr Ile Gly Val
420 425 430
Thr Ser Gly Ala Ser Thr Pro Asp Lys Val Val Glu Asp Ala Leu Gln
435 440 445
Lys Val Phe Glu Ile Lys Arg Gln Glu Ile Leu Gln Val Ala
450 455 460
<210> 10
<211> 397
<212> БЕЛОК
<213> Zea mays
<400> 10
Met Ala Ala Ser Gly Ala Trp Leu Ala Arg Ala Thr Ala Thr Ala Val
1 5 10 15
Leu Gly Phe Val Leu Ala Val Ala Ser Ala Glu Ala Ala Ser Gly Asp
20 25 30
Val Glu Met Val Phe Leu Lys Ala Ala Val Ala Lys Gly Ala Val Cys
35 40 45
Leu Asp Gly Ser Pro Pro Val Tyr His Phe Ser Pro Gly Ser Gly Ser
50 55 60
Gly Ala Asn Asn Trp Val Val His Met Glu Gly Gly Gly Trp Cys Arg
65 70 75 80
Asn Pro Asp Glu Cys Ala Val Arg Lys Gly Asn Phe Arg Gly Ser Ser
85 90 95
Lys Phe Met Lys Pro Leu Ser Phe Ser Gly Ile Leu Gly Gly Asn Gln
100 105 110
Lys Ser Asn Pro Asp Phe Tyr Asn Trp Asn Arg Val Lys Ile Arg Tyr
115 120 125
Cys Asp Gly Ser Ser Phe Thr Gly Asp Val Glu Ala Val Asp Thr Ala
130 135 140
Lys Asp Leu Arg Tyr Arg Gly Phe Arg Val Trp Arg Ala Val Ile Asp
145 150 155 160
Asp Leu Leu Thr Val Arg Gly Met Ser Lys Ala Gln Asn Ala Leu Leu
165 170 175
Ser Gly Cys Ser Ala Gly Gly Leu Ala Ala Ile Leu His Cys Asp Arg
180 185 190
Phe His Asp Leu Phe Pro Ala Lys Thr Lys Val Lys Cys Phe Ser Asp
195 200 205
Ala Gly Tyr Phe Phe Asp Gly Lys Asp Ile Ser Gly Asn Phe Tyr Ala
210 215 220
Arg Ser Ile Tyr Lys Ser Val Val Asn Leu His Gly Ser Ala Lys Asn
225 230 235 240
Leu Pro Ala Ser Cys Thr Ser Lys Pro Lys Gln Ser Pro Glu Leu Cys
245 250 255
Met Phe Pro Gln Tyr Val Val Pro Thr Met Arg Thr Pro Leu Phe Ile
260 265 270
Leu Asn Ala Ala Tyr Asp Ser Trp Gln Val Lys Asn Val Leu Ala Pro
275 280 285
Ser Pro Ala Asp Pro Lys Lys Thr Trp Ala Gln Cys Lys Leu Asp Ile
290 295 300
Lys Ser Cys Ser Ala Ser Gln Leu Thr Thr Leu Gln Asn Phe Arg Thr
305 310 315 320
Asp Phe Leu Ala Ala Leu Pro Lys Thr Gln Ser Val Gly Met Phe Ile
325 330 335
Asp Ser Cys Asn Ala His Cys Gln Ser Gly Ser Gln Asp Thr Trp Leu
340 345 350
Ala Asp Gly Ser Pro Thr Val Asn Lys Thr Gln Ile Gly Lys Ala Val
355 360 365
Gly Asp Trp Tyr Tyr Asp Arg Glu Val Pro Arg Gln Ile Asp Cys Pro
370 375 380
Tyr Pro Cys Asn Pro Thr Cys Lys Asn Arg Asp Asp Asp
385 390 395
<210> 11
<211> 311
<212> БЕЛОК
<213> Zea mays
<400> 11
Met Pro Lys Asp Arg Ser Ser Arg Val Ser Ser Tyr Glu Ser Arg Arg
1 5 10 15
Ala Gly Ala Ser Pro Tyr Phe Ser Ser Ser His Gly Gln Ser Ser Ser
20 25 30
Cys Arg Arg Ser Glu Glu Ser Cys Gly Ala Ala Ala Ala Ala Ala Ala
35 40 45
Lys Gln Ala Ala Glu Trp Glu Asp Val Arg Cys Pro Val Cys Met Asp
50 55 60
His Pro His Asn Ala Val Leu Leu Val Cys Ser Ser His Glu Lys Gly
65 70 75 80
Cys Arg Pro Phe Met Cys Asp Thr Ser Ser Arg His Ser Asn Cys Tyr
85 90 95
Asp Gln Tyr Arg Lys Ala Ser Lys Asp Ser Arg Thr Glu Cys Ser Glu
100 105 110
Cys Gln Gln Gln Val Gln Leu Ser Cys Pro Leu Cys Arg Gly Pro Val
115 120 125
Ser Asp Cys Ile Lys Asp Tyr Ser Ala Arg Arg Phe Met Asn Thr Lys
130 135 140
Val Arg Ser Cys Thr Thr Glu Ser Cys Glu Phe Arg Gly Ala Tyr Gln
145 150 155 160
Glu Leu Arg Lys His Ala Arg Val Glu His Pro Thr Gly Arg Pro Met
165 170 175
Glu Val Asp Pro Glu Arg Gln Arg Asp Trp Arg Arg Met Glu Gln Gln
180 185 190
Arg Asp Leu Gly Asp Leu Met Ser Met Leu Arg Ser Gly Phe Asn Ser
195 200 205
Asn Ile Glu Asp Asp Ser Gly Gly Leu Gly Asp Thr Glu Glu Gly Gly
210 215 220
Glu Glu Ala Glu Met Thr Pro Ala Ser Ile Thr Met Val Phe Ile Met
225 230 235 240
Pro Ser Arg Gly Ser Ile Met Gln Tyr Leu Ser Glu Arg Ser Arg Thr
245 250 255
Ile Ile Leu Val Ser Arg Arg Arg Ala Ser Ser Ser Ser Gly Gly Asp
260 265 270
Ala Glu Ala Thr Ala Pro Asp Ser Glu Glu Gly Asp Asp Pro Met Pro
275 280 285
Ser Ala Glu Ala Ser Ala Gly Ser Gln His Ser Ser Glu Gln Glu Glu
290 295 300
Ala Asp Gly Asp Pro Ala Gln
305 310
<210> 12
<211> 428
<212> БЕЛОК
<213> Zea mays
<400> 12
Met Ala Ala Ala Pro Thr Thr Val Thr Lys Ser Pro Pro Ser Leu Val
1 5 10 15
Pro Pro Ala Gly Pro Thr Pro Gly Gly Ser Leu Pro Leu Ser Ser Ile
20 25 30
Asp Lys Thr Ala Ala Val Arg Val Ser Val Asp Phe Ile Gln Val Phe
35 40 45
Pro Ala Pro Thr Ser Gly Lys Glu Asp Arg Ser Pro Ser Ser Thr Ile
50 55 60
Ala Ala Met Arg Glu Gly Phe Ala Lys Ala Leu Val Pro Tyr Tyr Pro
65 70 75 80
Val Ala Gly Arg Ile Ala Glu Pro Val Pro Gly Glu Pro Glu Ile Glu
85 90 95
Cys Thr Gly Glu Gly Val Trp Phe Val Glu Ala Glu Ala Ser Cys Ser
100 105 110
Leu Glu Glu Ala Arg Asn Leu Glu Arg Pro Leu Cys Ile Pro Lys Glu
115 120 125
Glu Leu Leu Pro Arg Pro Pro Ala Gly Val Arg Val Glu Asp Thr Leu
130 135 140
Leu Leu Ala Gln Val Thr Lys Phe Thr Cys Gly Gly Phe Ala Val Gly
145 150 155 160
Ile Cys Phe Ser His Leu Val Phe Asp Gly Gln Gly Ala Ala Gln Phe
165 170 175
Leu Lys Ala Val Gly Glu Met Ala Arg Gly Leu Pro Glu Pro Ser Ile
180 185 190
Lys Pro Ile Trp Ala Arg Asp Ala Ile Pro Asn Pro Pro Lys Pro Pro
195 200 205
Leu Gly Pro Pro Pro Ser Phe Thr Ala Phe Asn Phe Glu Lys Ser Val
210 215 220
Leu Glu Ile Ser Pro Asp Ser Ile Lys Asn Val Lys Asp Gln Val Ala
225 230 235 240
Ser Glu Thr Asn Gln Lys Cys Ser Thr Phe Asp Val Val Thr Ala Ile
245 250 255
Ile Phe Lys Cys Arg Ala Leu Ala Val Asp Phe Ala Pro Asp Ala Glu
260 265 270
Val Arg Leu Gly Phe Ala Ala Ser Thr Arg His Leu Leu Ser Asn Val
275 280 285
Leu Pro Ser Val Glu Gly Tyr Tyr Gly Asn Cys Val Tyr Pro Gly Gly
290 295 300
Leu Thr Lys Thr Ser Gln Glu Val Lys Glu Ala Ser Leu Val Glu Ile
305 310 315 320
Val Thr Val Ile Arg Glu Ala Lys Glu Ala Leu Ser Ser Arg Phe Leu
325 330 335
Asp Trp Leu Ser Gly Gly Ala Lys Glu Asn His Tyr Asn Val Ser Leu
340 345 350
Asp Tyr Gly Thr Leu Val Val Thr Asp Trp Ser His Val Gly Phe Asn
355 360 365
Glu Val Asp Tyr Gly Phe Gly Glu Pro Ser Tyr Val Phe Thr Leu Asn
370 375 380
Asp Asp Val Asn Ile Val Pro Ser Val Val Tyr Leu Lys Pro Pro Lys
385 390 395 400
Pro Lys Gln Gly Ile Arg Leu Val Leu Gln Cys Val Glu Gly His His
405 410 415
Ser Ala Val Phe Gly Glu Glu Leu Gln Lys His Ala
420 425
<210> 13
<211> 451
<212> БЕЛОК
<213> Zea mays
<400> 13
Met His Asp Leu Lys Lys Glu Leu Glu Gly Tyr Asn Thr Gly Asp Phe
1 5 10 15
Asp Glu Thr Asn Lys Lys Lys Ala Leu Asp Ala Leu Lys Arg Met Glu
20 25 30
Ser Trp Asn Leu Phe Arg Asp Thr Ser Val Glu His His Ser Tyr Thr
35 40 45
Val Ala His Asp Ser Phe Leu Ala Gln Leu Gly Ser Met Leu Trp Gly
50 55 60
Ser Met Arg His Val Ile Ala Pro Ser Ala Ser His Arg Val Tyr His
65 70 75 80
Tyr Tyr Glu Lys Leu Ser Phe Gln Leu Tyr Phe Val Thr Arg Glu Lys
85 90 95
Val Arg Ser Ile Lys Gln Leu Pro Val Asn Val Lys Ser Ile Arg Glu
100 105 110
Ser Leu Asn Ser Val Leu Leu His His Gln Asn Ser Met Phe Ser Gln
115 120 125
Asn Met Leu Ser Leu Ser Glu Asp Pro Ser Leu Met Met Ala Phe Ser
130 135 140
Met Ala Arg Arg Ala Ala Ala Val Pro Leu Leu Leu Val Asn Gly Thr
145 150 155 160
Tyr Lys Ser Thr Val Ser Thr Tyr Leu Asp Ser Ala Ile Leu Gln His
165 170 175
Gln Leu Gln Lys Leu Asn Glu His Asn Ser Leu Lys Gly Arg His Ser
180 185 190
Asn His Arg Ser Thr Leu Glu Val Pro Ile Phe Trp Phe Ile His Asn
195 200 205
Glu Pro Ile Leu Leu Asp Lys His Tyr Gln Ala Lys Ala Leu Ser Asn
210 215 220
Met Val Val Val Val Gln Ser Asp Asp Asp Ser Trp Glu Ser His Leu
225 230 235 240
Gln Cys Asn Gly Arg Pro Ile Leu Trp Asp Leu Arg Lys Pro Val Lys
245 250 255
Ala Ala Ile Ala Ala Thr Ala Glu Tyr Val Ser Gly Leu Leu Pro Pro
260 265 270
His Leu Val Tyr Ser His Ala His Glu Thr Ala Ile Glu Asp Trp Thr
275 280 285
Trp Ser Val Gly Cys Asn Pro Ser Ala Val Thr Ser Glu Gly Ser Gln
290 295 300
Leu Ser Glu Phe Gln Gln Asp Val Ile Ala Arg Asn Tyr Ile Ile Thr
305 310 315 320
Ser Val Glu Glu Ser Ile Gln Val Ile Asn Ser Ala Ile Gln Gln Leu
325 330 335
Val Ile Glu Arg Thr Thr Glu Lys Gly Phe Lys Ile Phe Lys Ala His
340 345 350
Glu Ser Lys Met Val Glu Lys Tyr Asn Ala Val Val Ser Leu Trp Arg
355 360 365
Arg Val Ser Ala Met Ser Lys Gly Leu Arg Tyr Gly Asp Ala Val Lys
370 375 380
Leu Met Ser Met Leu Glu Asp Ala Ser Asn Gly Phe Ser Ser Ala Val
385 390 395 400
Asn Ser Thr Ile Ser Ser Leu His Pro Val Gln Cys Thr Arg Glu Arg
405 410 415
Lys Val Asp Val Gln Leu Asp Leu Thr Thr Leu Pro Ala Phe Leu Ala
420 425 430
Val Phe Leu Leu Leu Trp Phe Leu Leu Arg Pro Arg Arg Pro Lys Pro
435 440 445
Lys Ile Asn
450
<210> 14
<211> 975
<212> БЕЛОК
<213> Zea mays
<400> 14
Met Lys Thr Arg Ile Val Tyr Ser Arg Glu Phe Leu Leu Ser Leu Gly
1 5 10 15
Glu Leu Glu His Cys Lys Lys Leu Pro Pro Asp Phe Asp Ala Ala Leu
20 25 30
Leu Ser Glu Leu Gln Glu Leu Ser Ala Gly Val Leu Glu Arg Asn Lys
35 40 45
Gly Tyr Tyr Asn Thr Ser Gln Gly Arg Pro Asp Gly Ser Val Gly Tyr
50 55 60
Thr Tyr Ser Ser Arg Gly Gly Asn Thr Gly Gly Arg Trp Asp Thr Arg
65 70 75 80
Ser Ser Gly Ser Ser Asp Arg Asp Gly Glu Pro Asp Arg Glu Ser Gln
85 90 95
Thr Gln Ala Gly Arg Gly Ala Asn Gln Tyr Arg Arg Asn Trp Gln Asn
100 105 110
Thr Glu His Asp Gly Leu Leu Gly Arg Gly Gly Phe Pro Arg Pro Ser
115 120 125
Gly Tyr Thr Gly Gln Leu Ser Ser Lys Asp His Gly Asn Ala Pro Gln
130 135 140
Leu Asn Arg Thr Ser Glu Arg Tyr Gln Pro Pro Arg Pro Tyr Lys Ala
145 150 155 160
Ala Pro Phe Ser Arg Lys Asp Ile Asp Ser Ile Asn Asp Glu Thr Phe
165 170 175
Gly Ser Ser Glu Leu Ser Asn Glu Asp Arg Ala Glu Glu Glu Arg Lys
180 185 190
Arg Arg Ala Ser Phe Glu Leu Met Arg Lys Glu Gln His Lys Ala Val
195 200 205
Leu Gly Lys Lys Ser Gly Pro Asp Ile Leu Lys Glu Asn Pro Ser Asp
210 215 220
Asp Ile Phe Ser Lys Leu Gln Thr Ser Thr Ala Lys Ala Asn Ala Lys
225 230 235 240
Thr Lys Asn Glu Lys Leu Asp Gly Ser Val Val Ser Ser Tyr Gln Glu
245 250 255
Asp Thr Thr Lys Pro Ser Ser Val Leu Leu Ala Pro Ala Ala Arg Pro
260 265 270
Leu Val Pro Pro Gly Phe Ala Asn Ala Phe Ala Asp Lys Lys Leu Gln
275 280 285
Ser Gln Ser Ser Asn Ile Thr His Glu Pro Lys Leu Glu Asp Asp Gln
290 295 300
Ser Ala Thr Gly Phe Thr Ser Glu Ser Lys Glu Lys Gly Val Ser Gly
305 310 315 320
Asn Asp Ala Thr Met Gly Pro Lys His Thr Leu Pro Pro Gly Ser Val
325 330 335
Thr Ser Ser Ala Glu Leu Ala Ser Ser Val Leu Lys Gly Ser Glu Asp
340 345 350
Trp Asp Ala Asp Val Met Asp Lys Tyr Ser Ile Gly Lys Glu Gly Lys
355 360 365
Ser Lys Asn Ile Asp Pro Val Arg Lys Asp Asp Ser Val Ala Ile Leu
370 375 380
Glu Gln Phe Phe Gly Asn Val Leu Ser Lys Ser Gly Ser Asn Leu Pro
385 390 395 400
Thr Tyr Val Glu Asn Gln Pro Leu Lys Thr Asp Asp Asp Met Ile Thr
405 410 415
Ser Val Pro Glu Ser Ser Lys Phe Ala His Trp Phe Leu Asp Glu Asp
420 425 430
Leu Lys Pro Ala Glu Asp Leu Ser Ser Lys Ser Leu Leu Ser Met Ile
435 440 445
Val Lys Asn Glu Asn Pro Gly Leu Glu Asn Leu Asn His Thr Pro Leu
450 455 460
Ser Asp Ala Ala Ala Gln Asn Leu Ser Pro Arg Ala Pro Ile Asp Lys
465 470 475 480
Leu Asp Ser Ala Ser Glu Leu Ile Ser Phe Thr Ser Ser Thr Pro Ala
485 490 495
Asn Gly Val Leu Glu Gln Cys Ile His Ser Asp Val Pro Glu Ala Val
500 505 510
Pro Ile Met Thr Cys Glu Asp Leu Glu Gln Thr Met Leu Ala Gln Val
515 520 525
Ser Asn Ser Ser Ser Thr Gln Ile Asn Ala Thr Lys Glu Gln Leu Thr
530 535 540
Val Met Asp Glu Pro Val Ala Met Gln Lys Val Thr Val Asp Asn His
545 550 555 560
Ala Ser Gln His Leu Leu Ser Leu Leu Gln Lys Gly Thr Asp Asn Lys
565 570 575
Gly Ala Pro Ser Leu Gly Phe Gln Arg Glu Ser Thr Asp Glu Pro Leu
580 585 590
Ser Val Asp Thr Asn Leu Met Ala Asn Gly Gly Ile Ser Gly Ser Asp
595 600 605
Pro Val Asn Ser Val Glu Asn Val Pro Thr Ser Gly Lys Asp Leu Thr
610 615 620
Leu Glu Ala Leu Phe Gly Ala Ala Phe Met Asn Glu Leu His Ser Lys
625 630 635 640
Asp Ala Pro Val Ser Ile Arg Gly Ala Thr Thr Gly Gly Pro Thr Glu
645 650 655
Phe Ala Glu Met Gly Lys Thr Leu Leu Ser Ser Ser His Glu Gly Tyr
660 665 670
Tyr Pro Val Glu Gln Thr Val His Phe Asn Asn Thr Lys Asp Ala Ala
675 680 685
Val Arg Arg Glu Pro Gly Ile Glu His Ser Ala Val Pro Gly Leu Ser
690 695 700
Gln Gly Ser Ala Ser Phe Asp Lys Lys Gly Met Glu Ile His Leu Pro
705 710 715 720
Glu Glu Asp Asn Leu Phe Thr Met Ser Asp Ser Leu Leu Gly Gln Asn
725 730 735
Ser Asp Ile Leu Ala Ser Val Gly Ser Ser Arg Val Glu Gly Leu Leu
740 745 750
Pro Glu Lys Ala Leu Asp Asn Leu Ser Tyr Arg Phe Gln Ser Leu Val
755 760 765
Pro Gly Asp Ala Glu His Ile Gln Val Tyr Gly Pro Asp Ala Leu Gly
770 775 780
Ser His Pro Arg Asp Ser Gln Asn Met Tyr His Leu Leu Gln Gly Arg
785 790 795 800
Pro Pro Met Ile Ala Pro His Pro Met Met Asp His Ile Val Asn Arg
805 810 815
Lys Gln Pro Ala Pro Phe Asp Met Ala Gln Ser Ile His His Asp Ser
820 825 830
His Arg Ser Phe Pro Ser Asn Val Asn His Met Gln His Asn Leu His
835 840 845
Gly Pro Gly Val Pro His Leu Asp Pro Ala Gly His Ile Met Arg Gln
850 855 860
His Met Ser Met Pro Gly Arg Phe Pro Pro Glu Gly Leu Pro Arg Gly
865 870 875 880
Val Pro Pro Ser Gln Pro Val His His Met Ala Gly Tyr Arg Pro Glu
885 890 895
Met Gly Asn Val Asn Asn Phe His Met His Pro Arg Gln Pro Asn Tyr
900 905 910
Gly Glu Phe Gly Leu Met Met Pro Gly Pro Glu Val Arg Gly Asn His
915 920 925
Pro Glu Ala Phe Glu Arg Leu Ile Gln Met Glu Met Ser Ala Arg Ser
930 935 940
Lys Gln Gln Gln Val His His Pro Ala Met Ala Ala Gly Arg Val Pro
945 950 955 960
Ser Gly Met Tyr Gly His Glu Leu Asp Ala Lys Leu Arg Tyr Arg
965 970 975
<210> 15
<211> 401
<212> БЕЛОК
<213> Zea mays
<400> 15
Met Ala Ser Leu Ala Gln His Val Ala Gly Leu Pro Cys Pro Pro Leu
1 5 10 15
Ser Gly Ala Ser Arg Arg Arg Pro Ala Ala Gln Arg Arg Pro Pro Ser
20 25 30
Ala Leu Val Cys Gly Thr Tyr Ala Leu Thr Lys Asp Glu Arg Glu Arg
35 40 45
Glu Arg Met Arg Gln Val Phe Asp Asp Ala Ser Glu Arg Cys Arg Thr
50 55 60
Ala Pro Met Glu Gly Val Ala Phe Ser Pro Asp Asp Leu Asp Thr Ala
65 70 75 80
Val Glu Ser Thr Asp Ile Asp Thr Glu Ile Gly Ser Leu Ile Lys Gly
85 90 95
Thr Val Phe Met Thr Thr Ser Asn Gly Ala Tyr Ile Asp Ile Gln Ser
100 105 110
Lys Ser Thr Ala Phe Leu Pro Leu Asp Glu Ala Cys Leu Leu Asp Ile
115 120 125
Asp Asn Val Glu Glu Ala Gly Ile Arg Pro Gly Leu Val Glu Glu Phe
130 135 140
Met Ile Ile Asp Glu Asn Pro Gly Asp Glu Thr Leu Ile Leu Ser Leu
145 150 155 160
Gln Ala Ile Gln Gln Glu Leu Ala Trp Glu Arg Cys Arg Gln Leu Gln
165 170 175
Ala Glu Asp Val Val Val Thr Gly Lys Val Ile Gly Gly Asn Lys Gly
180 185 190
Gly Val Val Ala Leu Val Asp Gly Leu Lys Gly Phe Val Pro Phe Ser
195 200 205
Gln Val Ser Ser Lys Thr Thr Ala Glu Glu Leu Leu Glu Lys Glu Leu
210 215 220
Pro Leu Lys Phe Val Glu Val Asp Glu Glu Gln Gly Arg Leu Val Leu
225 230 235 240
Ser Asn Arg Lys Ala Met Ala Asp Ser Gln Ala Gln Leu Gly Ile Gly
245 250 255
Ser Val Val Leu Gly Thr Val Glu Ser Leu Lys Pro Tyr Gly Ala Phe
260 265 270
Ile Asp Ile Gly Gly Ile Asn Gly Leu Leu His Val Ser Gln Ile Ser
275 280 285
His Asp Arg Val Ala Asp Ile Ser Thr Val Leu Gln Pro Gly Asp Thr
290 295 300
Leu Lys Val Met Ile Leu Ser His Asp Arg Glu Arg Gly Arg Val Ser
305 310 315 320
Leu Ser Thr Lys Lys Leu Glu Pro Thr Pro Gly Asp Met Ile Arg Asn
325 330 335
Pro Lys Leu Val Phe Glu Lys Ala Asp Glu Met Ala Gln Ile Phe Arg
340 345 350
Gln Arg Ile Ala Gln Ala Glu Ala Met Ala Arg Ala Asp Met Leu Arg
355 360 365
Phe Gln Pro Glu Ser Gly Leu Thr Leu Ser Ser Glu Gly Ile Leu Gly
370 375 380
Pro Leu Ser Ser Asp Ala Pro Ser Glu Asp Ser Glu Asp Arg Thr Asp
385 390 395 400
Glu
<210> 16
<211> 279
<212> БЕЛОК
<213> Zea mays
<400> 16
Met Gly Ser Gly Val Ser Ser Ser Met Ala Leu Ala Leu Ala Gly Phe
1 5 10 15
Cys Phe Ser Val Leu Phe Ile Val Phe Val Cys Thr Arg Leu Ala Cys
20 25 30
Ala Leu Val Arg Arg Arg Arg Arg Gln Ala Arg Ala Arg Leu Ala Ala
35 40 45
Ala Pro Pro Leu Pro His Tyr Ala His Gly Tyr Ala Asp Pro Asp Pro
50 55 60
Phe Pro Ser Phe Arg Ala Ala Arg His His His His Ala Pro Gly Leu
65 70 75 80
Asp Pro Ala Ala Phe Pro Thr Arg Ala Tyr Ala Ala Ala Gln Ala Ser
85 90 95
Asp Ser Asp Asp Gly Ser Gln Cys Val Ile Cys Leu Ala Glu Tyr Glu
100 105 110
Glu Gly Asp Glu Leu Arg Val Leu Pro Pro Cys Ser His Thr Phe His
115 120 125
Thr Gly Cys Ile Ser Leu Trp Leu Ala Gln Asn Ser Thr Cys Pro Val
130 135 140
Cys Arg Val Ser Leu Leu Val Pro Asp Thr Ser Thr Thr Pro Glu Ser
145 150 155 160
Glu His Ser Ala Pro His Pro Pro Pro Pro Pro His His His His His
165 170 175
Leu Ser Ser Ile Val Ile Ile Ser Pro Pro Ser Ser Pro Glu Pro Ser
180 185 190
Arg Ser Asp Pro Cys Arg Cys Leu Phe Ala Ser Gly Gly Gly His Ser
195 200 205
Ser Arg Ala Ala Glu Ala Pro Pro Pro Pro Pro Pro Pro Arg His Glu
210 215 220
Pro Asp Gln Val Val Ser Gly Pro Pro Pro Ala Ala Asp Gly Ala Ser
225 230 235 240
Gly Tyr Ser Ser Pro Leu Pro Glu Val Ile His Pro Ala Pro Ala Pro
245 250 255
Glu Thr Asn Gly Gln Thr Val Arg Lys Gln Ala Gly Ser Arg Ser Thr
260 265 270
Thr Pro Leu Gly Pro Cys Lys
275
<210> 17
<211> 801
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (26)..(31)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (174)..(174)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (260)..(260)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (790)..(790)
<223> n представляет собой a, c, g или t
<400> 17
caagcaagct gtgtagraac aaacannnnn ngttaaatca gtacagctcy aagtcacctt 60
atctggagtt gaggcacctg aagtaacacc aatggtaata ggcccctcgg gtaaccagtt 120
atttttctca accagttcac catgctgtta aacaatccaa ttttcggtta aaantgagat 180
catatacttt caataawtaa atttaagctc tttccagttt ctaattaaaa gaaggtccct 240
tcaaaattct attatttttn aaaaaatgaa tgctgtggaa ggtatgagaa attacattta 300
rcttgtagct gatcctgttt cctggtccaa tcctttgttc actgtcaatc cagtatgatg 360
gaattccact gagttctccr atttcttgca gatgagaggt gttacttgaa ttccatcctc 420
caacaacaag aataaggtca actttctctt tcaccagctg atacatagca tcttgtcttt 480
cctgaggaat caaacatatg gtcggatgta tataagtcaa tgcagcatat gtcaaaacaa 540
ttataaatct atcacycaaa gagwcawttt ctcaaaccaa aagtggagca acaaatgkac 600
taggatcctc ttaactttga tccmaaataa aagtgccatc tagttgyctc agtttccttt 660
gcaggttgca tgtaaagggt gatggagatt gtattatacc atgaatggct accrtctatc 720
atgaattgta ygctagcctt ttctwgacrt tacagtacag tagtgctaaa tccatcattc 780
ccagtttttn aaatcatcaa g 801
<210> 18
<211> 801
<212> ДНК
<213> Zea mays
<400> 18
cggtggggga ctggtactac gatagggagg tccctcggca gattgattgc ccgtatccct 60
gcaacccaac ttgcaagaac cgtgatgatg attgagcaat tgtataagta gttcatgtta 120
tcgaaatgaa aacaataaag gatcacaacg cgcgcccgta gttgtagatg atgaattata 180
aacacatatg actgagctca aagttgttta atcatcatct gttgcgaaat gaggaagaca 240
attggtgtct tgaagctgtg ttttcgactg tgtctaaagc gtaaatgtaa cgtayattgt 300
gtcttsgcct atgcttaaga catkggacta gttgattggt caatttaatt tattaaatgt 360
tttgattggt gtaatgaata taataagtcg tgcatgccgc gtgactaggc ttccagtctt 420
ccacttacac cggctaagca ctgtctatat atatgtartc actttggatc aatgaatcag 480
ctgtttttat cagtttaggt tttctttttc acttcttgtt ttgccatggc tgagactggc 540
cgcagcgctt cccgccagta gtcctctcct ctatcactgt tcctgtttag cgtcactcta 600
ctgctgcgtc agtcgtctct acacttgcct ctacgcgcta cagctctaga ggaatacata 660
ggaccacgct agatgtggcg ggctgcaagc tgtcccctgc cctcgaaacc agtgcacgac 720
gtgggccctc ttctaattgt tagtataaga aaaattgata atagataaaa aatagtatat 780
gaaatgatat tttatggttg t 801
<210> 19
<211> 801
<212> ДНК
<213> Zea mays
<400> 19
agagtttgga gtaaagaaac caaccctgca tctgtattct gtctgtctgt gctgcttcga 60
ataagccttg catctcgctg acttgrgata taactatgcc gaaggacagg agctcccgcg 120
tttcctctta ygagagccgc cgggctggtg cctccccata cttctcatcg tctcatggac 180
agagcagttc ttstcgccgg tcmgaggagt cttgtgkggc agcagcggcg gcrgcagcaa 240
agcaagctgc agagtgggag gaygttcggt gcccggtgtg catggaccac ccgcacaacg 300
ccgtcctgct ggtctgctcc tcacacgaga agggctgccg ccccttcatg tgcgacacca 360
gctcgcggca ctcgaactgc tatgaccagt accggaaggc atccaaggat tcaaggacag 420
agtgcagcga gtgccagcag caggttcagc tctcgtgccc actgtgccgt gggccggtca 480
gcgattgcat caaggactac agcgcgcgga ggttcatgaa caccaaggtc cggtcgtgca 540
ccacggagtc gtgcgagttc aggggcgcct accakgagct gaggaagcat gctagggtgg 600
agcatccaac aggaaggcca atggaggtag accctgagcg gcagcgggac tggcgccgga 660
tggagcagca acgggacctt ggrgacttga tgagcatgct gcgttcaggg ttcamcagca 720
atattgagga cgacagtggc gggcttggag acaccgaaga agggggagag gaagctgaaa 780
tgactccggc ctccataacc a 801
<210> 20
<211> 801
<212> ДНК
<213> Zea mays
<400> 20
caggccaacg artctgaaat aatttgttcc aaatgggaac attattattc yttcgtcact 60
gtcaatattc atcacatgta taatgtaacg tgctgataat ggtacaaagt ataaacacga 120
ccaatctgcc ttattgsaag caawttccgg gagcagcaat ttgacacaac aaataaaata 180
caacagattc ccactgtrga atacactcac tctatgcatg cttctgcaac tcctcgcyga 240
acacrgcaga gtgatggcct tccacgcact gcagsaccag cctgatgccc tgcttcggct 300
tgggcggctt caggtayaca acggagggga cgatgttcac gtcgtcgttc agggtgaaca 360
cgtagctcgg ctcaccgaac ccgtagtcva cctcgttgaa acccacatgg ctccagtcag 420
tcacgacgag ggtgccrtag tctagcgaca crttgtagtg gttctccttg gcgccgccgc 480
tcaaccagtc aaggaaccty gaygacagag cttccttggc ttccctgatc rcggtcacra 540
tctccacaag cgaagcttcc ttcacctcct ggctggtctt ggtgagacca cctgggtaca 600
cacagttccc gtagtagcct tcgacygagg gcagcacatt gctcagcagg tggcgagtgc 660
tggctgcgaa gcccaagcgr acctcagcgt cgggcgygaa gtcgactgcc aaggcgcggc 720
atttgaagat tatggcmgtg accacgtcga aagtggaaca cttctggttg gtttcactyg 780
caacctgatc cttcacrytc t 801
<210> 21
<211> 801
<212> ДНК
<213> Zea mays
<400> 21
ggccgggaat tgttccgacc caccaatccg acacaaacga acaaggcctt acccggaacg 60
aggaagaatt ggctaaaccc ggaacgagga caggactgra tgaattaaag ttttcatgac 120
attccaactc tgctacaact tggggawgtg tcaggattgy aatcttctga aaccttgttc 180
cctgccaccc ggtgttttgg acctttggaa ttcccgrgcc atgctccagc gccatctgta 240
atccatgcgc gtattcacaa aaastctatg gcctatgctg ctcattggct cggtgttcag 300
ttgatcttag gcttcggtct ccttggacgt agaagaaacc aaagcaacaa aaatacagct 360
agaaaagcag gaagtgttgt caagtctagc tgcacgtcga gcttcctttc gcgggtgcat 420
tggacagggt gcagacttga aatggtggag ttcacagcac tagaaaacct gaaaagaagt 480
aacactataa ttcagtcaaa gaagtacaat gaatcagggg ccctattcta gtcgcagata 540
taactatgtt ttgttttcta agtcggacaa cttgctggtt gttatgattg accgacttgg 600
gcgattaatc acgattagtc ggacgacttg ggcgattaat cttacgactt gaaaacagta 660
tactatgttc acccggtaaa aaacctcatc ccacgacagg attatctccc ccaaggcatg 720
catgtatgag gagcggtaac cagygcggca cwgcctatga ctggatctta acggatacaa 780
gcacacagtg aagggttaga a 801
<210> 22
<211> 801
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (662)..(662)
<223> n представляет собой a, c, g или t
<400> 22
aaaaggcact tgataacctc agctataggt ttcaargtct tgtgcctggt gatgcagaac 60
acattcaagt atatggtcct gatgcacttg gatctcatcc tcgtgattct cagaatatgt 120
atcatcttct acagggtagg cctcctatga trgcacctca ccctatgatg gatcacattg 180
ttaataggaa acagccagct ccatttgata tggcacagtc gatacaccat gattctcacc 240
gttctttccc atctaatgtg aatcatatgc aacataatct tcatgggcca ggggtccctc 300
acttggaccc tgctggacat attatgcgac aacacatgtc catgcctgga agatttcctc 360
cagaaggctt gccaagaggt gtccctccat ctcagcctgt ccatcacatg gctggttata 420
gacctgaaat gggtaatgta aataatttcc atatgcaccc tcgccagccc aactatggag 480
aatttggatt gatgatgcca ggcaagtctc aattgtccta attctatttg ttctattaac 540
tggcagatta ctttgtcatt atttgcaagt tcagacatgc catagtgcca gactttctat 600
cggtggcact gttaacttat catactccct ccgtcccaaa atatagttct ttctagctca 660
cntttttttt ctgtccacat tcttttaaat gataattaat atagatatac atgtaaactg 720
cgttcatatg ttacttaata aatgtgtgat tagtctwaaa aaattatatt ttrggatgga 780
gggagtactg gttttgcata t 801
<210> 23
<211> 801
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (787)..(787)
<223> n представляет собой a, c, g или t
<400> 23
gtagaaaggc tgactcggcc tctttcacgg tcatggctca gtatcataac ctataagcaa 60
cakrattcat tggtgttagc tccattaagt atggatatgg cataacaacc aagaaccaca 120
gccacaatac cttgagggta tctcctggtt gcagaactgt tgagatatct gcaacacggt 180
catgactaat ctggctcaca tggagaaggc cgttgattcc rccgatgtcr atgaaggcrc 240
cataaggttt taggctctcw acagttccca agacaactga tccaatwcct agctgggcct 300
gactatctgc cattgccttg cgattactga ggacaagcct gccttgttcc tcatcgacct 360
ctacaaactt cagaggcaat tctttctcaa gcagctcttc agcggttgtt ttctgatcaa 420
gatcaaacaa aagtttatca aataaggaat tgtaaagctc agctagagca aagcatcaaa 480
cataaaatat ggtaaataya tgaaaggcag ctcatgcttg ccaattaaac gtagaatata 540
agaaacttct gtgaaaagat caagaactaa ccgatgacac ttgcgaaaat ggaacgaaac 600
ccttaagccc atccacaaga gctacwacac ctcctttgtt tccaccaatt acctgcaagc 660
aaaattgaat aaggttraag aaactaagat aacctagaaa ggctgaacaa tgacataaag 720
gtattcccaa cagagaccag ccatatataa agttatacct tcccctactg ttttatggta 780
aaaaaancac tataaacaaa t 801
<210> 24
<211> 801
<212> ДНК
<213> Zea mays
<400> 24
aggaaatggc tacatgattt ctcactaaca ttaagcacag ggttggggat ataaatacct 60
aacacattgt acyagtagag attcaggcaa tatgttggta ctagctaacg gtgcgtgaaa 120
ttggctttga ggtgtccaag gaaacttgca tatgacgaga accaccgtag aacttgtatt 180
agcctatagc actactgtct acagtgtcca accaacttgt gawtgaaaac aactacatta 240
agaaccttag cctcgcgccc agaatagmga gacctctgca tcagtgatta ccctctatac 300
ctgtactctc tgagagagca ccaaacgagc atgacagcga tatccggaag agcccaatta 360
ttcccagtga acagcgcgag cgaccccggg atgaagcaga ccaccccatt tccaatctgg 420
ttccgtgcac cccccgsaat agatccccgg cggtggaccc ggcccctcct tcccygcaaa 480
cctcctccga gggccggccg gccttcgcag ctacgctagt tgagtcggca tcgggatccg 540
caaaaccacc aacgaatcct gccggcagaa acgtgcgcga tcgcgtgacg gcggcacagc 600
ctgcgactgc aagtctgaaa tcggtggggt gggtacctag tggcgatggc gagagctagc 660
ccaccmcccg cgccggcaag cggtagcagg ctccagttgt tgtactgtac ccacagcaac 720
gggcaactcc accgaaccgc acgcgaaagc aggagatcgg tgggggtgga gcgggggtgg 780
ggagggtggg ttggattcga a 801
<210> 25
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 25
tgttcactgt caatccagta tgatg 25
<210> 26
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 26
cctcaggaaa gacaagatgc tatg 24
<210> 27
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 27
tgcagatgag aggtgttact 20
<210> 28
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 28
ttgcagatga gaggtattac tt 22
<210> 29
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 29
tagccggtgt aagtggaaga 20
<210> 30
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 30
tcgactgtgt ctaaagcgta aatg 24
<210> 31
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 31
cctagtcatg cggcat 16
<210> 32
<211> 15
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 32
ctagtcacgc ggcat 15
<210> 33
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 33
tcgctgcact ctgtcctt 18
<210> 34
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 34
cggcactcga actgctatga c 21
<210> 35
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 35
atccttggat gccttcc 17
<210> 36
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 36
aatccttgga ggccttc 17
<210> 37
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 37
gctcaccgaa cccgtagtc 19
<210> 38
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 38
gcgccaagga gaaccac 17
<210> 39
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 39
tcgttgaagc ccacat 16
<210> 40
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 40
tcgttgaaac ccacatg 17
<210> 41
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 41
gcaggaagtg ttgtcaagtc ta 22
<210> 42
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 42
agtgctgtga actccaccat 20
<210> 43
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 43
cacgtcgagc ttcctt 16
<210> 44
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 44
cacgtcgacc ttcctt 16
<210> 45
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 45
ccaagaggtg tccctccatc t 21
<210> 46
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 46
gacttgcctg gcatcatcaa tc 22
<210> 47
<211> 15
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 47
cagcctgtcc atcac 15
<210> 48
<211> 15
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 48
cagcctgtgc atcac 15
<210> 49
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 49
gttcctcatc gacctctaca aac 23
<210> 50
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 50
gctctagctg agctttacaa ttcct 25
<210> 51
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 51
agctcttcag cggttgt 17
<210> 52
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 52
agctcttcgg cggttg 16
<210> 53
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 53
gagcccaatt attcccagtg aacag 25
<210> 54
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ПРАЙМЕР
<400> 54
gggtgcacgg aaccagat 18
<210> 55
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 55
aagcagagca ccccat 16
<210> 56
<211> 16
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ЗОНД
<400> 56
aagcagacca ccccat 16
<210> 57
<211> 801
<212> ДНК
<213> Zea mays
<400> 57
tacaagcaag ytgtgtagra acaaacastt waagttarat cagtacagct cyaagtcacc 60
ttrtctggag ttgaggcacc tgaagtaaca ccaatrgtaa taggcccctc gggtaaccag 120
ttatttttct caacmagttc accatgctgt taaacaatcc aattttcrgt taaaatgaga 180
tcatatactt tcaataawta aatttaagct ctttccagtt tmtaaytaaa agaakgtccc 240
ttcaaaattc tattattttt aaaaaatgaa tgctgtkrar ggtatkagaa attacrttta 300
rcttgtagct gatcctgttt cctggtccaa tcctttgttc actgtcaatc cagtatgatg 360
gaattccact gagttctccr atttcttgca gatgagargt rttacttgaa ttccatcctc 420
caacaacaag aataaggtca actttctcwt tcaccagctg atacatagca tcttgtcttt 480
cctragraat caaacatatr gtyrkatgta tataagtcaa tgcagcatat gtcaaaacaa 540
ttataaayct rtmacycaaa gagwcawttt ctcaaaccaa aagtggagma acaaatgkay 600
taggatcctc ttarctttgr tccmaaataa aagtgycatc tagttgyctc agtttcctty 660
gcaggttgca tgtaaagggt ratggagatt gtattatacc atgaatggct accrtctatc 720
rtgaattgta ykctagcctt ttctwgacrt tacagtacag tagtgctaaa tccatcattc 780
ccagttttta aatcatcaag a 801
<210> 58
<211> 801
<212> ДНК
<213> Zea mays
<400> 58
acaaccataa aatatcattt catataytat tttttatcta ttatcaattt ttcttmtact 60
aacaattaga agagkkccca ygtcgtgcac tggtttcgag ggcaggggac agcttgcagc 120
ccgccacatc tascgtggtc ctatgtattc ctctagagct gtagcgckta gagrcaagtg 180
tagagacgac tgacgcagca gtaragtgay gctaaacagr aacagtgrta gaggagagga 240
ytactggcgg gaagcgctgc ggccagtctc agccatggca aaacaagaag tgaaaaagar 300
aacmtaaact gataaaaaca gctgattcat tgatccaaag tgastacaya tatatakaca 360
gtgcttagcc ggtgtragtg gaagactggr agcctagtca ygcrrmatgc acracttatt 420
atattcatta caycratyaa aayatttaat aaattaaawt raycaatsaa ctartcymat 480
gycttaagya tagrcsaaga cacartrtay gttacattta cgctttagac ayagtcraaa 540
acayagcttc aagacaccra ttgtcttyct catttykcaa cmgakgatga ttaaacaact 600
ttgagctcag tcatatgtgt ttataattca tcatctacaa ctacgsgcgc gcgttgtgat 660
cctttattgt tttcatttcg ataacrtgaa ctacttatac wwttrctcaa tcatcatcac 720
ggttcttgca agttgggttg cagggatacg ggcaatcaat ctgccgaggg acctccctat 780
cgwagtacca gtcccccacy g 801
<210> 59
<211> 801
<212> ДНК
<213> Zea mays
<400> 59
tggttatgga ggccggagtc atttcagctt cctctccccc ttcttcggtg tctccaagcc 60
cgccactgyc gtcctcaaya ytgctgbtga accctgaacg cagcatgctc atcaagtcyc 120
caaggtcycg ttgctgctcc atccggcgcc artcccgctg ccgctcaggg tctacctcca 180
ttggccttcc tgttggatgc tccaccctag catgcttcct cagctcmtgg targcgcccc 240
tgaactcgca cgactccgtg gtgcacgacc ggaccttggt gttcatgaac ctccgcgcgc 300
tgtagtcctt gatgcaatcg ctgaccggcc cacggcacag tgggcacgag agctkaacct 360
gctgctggca ctcgctgcac tctgtmcttg aatccttgga kgccttccgg tactggtcat 420
agcagttcga gtgccgcgag ctggtgtcgc acatgaaggg gcggcagccc ttctcgtgtg 480
aggagcagac cagcaggacg gcgttgtgcg ggtggtccat gcacaccggr caccgaacrt 540
cctcccactc tgcagcttgc tttgctgcyg ccgccgctgc tgccccacaa gactcctckg 600
accggcgasa agaactgctc tgtccatgmg acgatgagaa gtatggrgag gcaccagccc 660
ggcggctctc rtaagaggaa acgcgggagc tcctgtcctt cggcatagtt atatcycaag 720
tcagcgagat gcaaggctta ttcgaagcag cacagacaga cagaatacag atgcagggtt 780
ggtttcttta ctccaaactc t 801
<210> 60
<211> 801
<212> ДНК
<213> Zea mays
<400> 60
caggccaacg artctgaaat aatttgttcc aaatgggaac aktattattc yttcgtcact 60
gtcaatattc atcacatgta taatgtaacg tgcygataat ggtacaaagt ataaacayga 120
ccaatmtgmc ttattgsaag caawttccrg gagcagcaat ttgacrcaac aaayaaaata 180
caacasattc ccastgtwga atacactcac tctatgcatg cttctgcaac tcctcgcyga 240
acacrgcaga gtgatggcct tccacgcact gcagsaccag cctgatgccc tgcttcggct 300
tgggcggctt caggtayaca acggagggga cgatgttcac gtcgtcgttc agggtgaaca 360
crtagctcgg ctcaccgaac ccgtagtcma cctcgttgaa rcccacatgg ctccagtcag 420
tcacgacgag ggtgccrtag tctarcgaca crttgtagtg rttctccttg gcgccgccgc 480
tcaaccagtc aaggaaccty gaygacagag cttccttggc ttccctgatc ryggtcacra 540
tctccacaag cgaagcttcc ttcacctcct ggctggtctt ggtgasacca cctgggtaca 600
cacagttccc gtagtagcct tcgacygagg gcagcacrtt gcycagcagg tggcgagtgc 660
tggctgcgaa gcccarrcgr acctcagcrt cgggygygaa gtcgaytgcc awggcrcggc 720
atttgaagat tatggcmgtg accacrtcga aagtggaaca cttctggttg gtttcactyg 780
caacctgatc cttcacrytc t 801
<210> 61
<211> 801
<212> ДНК
<213> Zea mays
<400> 61
ttctaaccct tcactrtgtg yttgtatccg ttaagatcca gtcataggcw gtgccgcrct 60
ggttaccgct cctcatacat gcwtgccttg ggggagatra tccygtcgtg ggatgaggtt 120
ttttaccggg tgaacatagt atactgtttt caagtcgtmw gattaatcgy ccargtcgtm 180
cgactaatcg tgattaatcg cccaagtcgg tcaatcataa caaccagcaa gtygtccgac 240
ttagaaaaca aarcatagtt atatctgcga ctagaatagg rcccctgatt cattgtactt 300
ctttkmctga attatagtgt tacwwctttt caggttttct agtgctgtga actccaccat 360
ttcaagtctr caccctgtcc aatgcacccg sgaaaggaag stcgacgtgc agctagactt 420
gacaacactt cctgcttttc tagctgtatt tttgttgctt tggtttcttc tacgtccaag 480
gagaccgaag cctaagatca actgaacacc gagccaatga gcagcatagg ccatagastt 540
tttgtgaatr cgmgcatgga ttacagatgg cgctggagca tggcycggga attccaaagg 600
tccaaaacac cgggtggcag ggaacaaggt ttcagaagat trcaatcctg acacwtcccc 660
aagttgtagy agagttggaa tgtcatgaaa actttaatty atycagtcct gtccycgttc 720
crggtttagc caattcttcc tcgttccggg taaggccttg ttcgtttgtg tcggattggt 780
gggtcggaac aattyccgrc c 801
<210> 62
<211> 801
<212> ДНК
<213> Zea mays
<400> 62
atatgcaaaa ccagtactcc mtccrtccya aaatataatt ttttwagact aatcacacat 60
ttattaagta acatatgaac gcagtttaca tgtatatcta tattaattat catttaaaag 120
aatgtggaca gaaaaaaaar gtragctaga aagaactata ttttgggayr gagggagtat 180
rrtaagttaa cagtgccacy gatagaaagk ctggcactat ggcatgtctg aacttgcaaa 240
taatgacaaa gtaatctgcc agttaataga acwwatagaa ttwrgacaat tgagacttgc 300
ctggcatcat caatccaaat tctccatagt tgggctggcg agggtgcata tggaaattat 360
ttacattacc catytcaggt ctataaccag ccatgtgatg sayaggctga gatggagrga 420
cacctcttgg caagccttct ggaggaaatc ttccaggcat ggacatgtgt tgtcgcataa 480
tatgtccagc agggtccaag tgagggaccc ctggcccatg aagattatgt tgcatatgat 540
tyacattaga tgggaaagaa cggtgagaat catggtgtat cgaytgtgcc atatcaaatg 600
gagctggytg tttcctatta acaatgtgrt ccatcatagg gtgaggtgcy atcataggag 660
gcctaccctg tagaagatga tacatattct gagaatcacg aggatgagat ccaagtgcat 720
caggaccawr tacttgaatg tgttctgcat caccaggcac aagacyttga aacctatarc 780
tgaggtyatc aagtgccttt t 801
<210> 63
<211> 1001
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (103)..(103)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (264)..(264)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (309)..(309)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (321)..(321)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (330)..(330)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (339)..(339)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (360)..(360)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (387)..(387)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (414)..(414)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (429)..(429)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (675)..(675)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (696)..(696)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (717)..(717)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (726)..(726)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (741)..(741)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (907)..(907)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (909)..(909)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (991)..(991)
<223> n представляет собой a, c, g или t
<400> 63
ttcagcacat gagaaaatgt tgcttttgtt tattcttacc ttctcaaaca caagcttggg 60
attgcggatc atgtcaccag gtgttggctc aagcttctta gtngaaaggc tgactcggcc 120
tctttcacgg tcatggctca gtatcataac ctataagcaa cagaattcat tggtgttagc 180
tccattaagt atggatatgg cataacaacc aagaaccaca gccacaatac cttgagggta 240
tctcctggtt gcagaactgt tganatatct gcaacacggt catgactaat ctggctcaca 300
tggagaagnc cgttgattcc nccgatgtcn atgaaggcnc cataaggttt taggctctcn 360
acagttccca agacaactga tccaatncct agctgggcct gactatctgc catngccttg 420
cgattactna ggacaagcct gccttgttcc tcatcgacct ctacaaactt cagaggcaat 480
tctttctcaa gcagctcttc rgcggttgtt ttctgatcaa gatcaaacaa aagtttatca 540
aataaggaat tgtaaagctc agctagagca aagcatcaaa cataaaatat ggtaaatata 600
tgaaaggcag ctcatgcttg ccaattaaac gtagaatata agaaacttct gtgaaaagat 660
caagaactaa ccgangacac ttgcgaaaat ggaacnaaac ccttaagccc atccacnaga 720
gctacnacac ctcctttgtt nccaccaatt acctgcaagc aaaattgaat aaggttaaag 780
aaactaagat aacctagaaa ggctgaacaa tgacataaag gtattcccaa cagagaccag 840
ccatatataa agttatacct tcccctactg ttttatggta aaaaaacact ataaacaaat 900
agacaanana ggagcaaaga tatataacac cttgaagaca gatgacatgt tctcataaac 960
tgactgatcc taacttcata agttcaataa ntgtagcaca t 1001
<210> 64
<211> 1001
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (3)..(3)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (47)..(47)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (88)..(88)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (90)..(90)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (104)..(104)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (107)..(107)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (112)..(112)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (125)..(125)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (225)..(225)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (241)..(241)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (260)..(260)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (264)..(264)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (266)..(266)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (317)..(317)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (319)..(319)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (368)..(368)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (445)..(445)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (987)..(987)
<223> n представляет собой a, c, g или t
<400> 64
canagggctt catgatttgt cagtaacact gagcaagaag aacaggnatg tcaaatggct 60
tcatgatttc tcaggaacac caatagcnan gcacaggctt aggnaanggc tncatgattt 120
ctcantaaca ttaagcacag ggttggggat ataaatacct aacacattgt accagtagag 180
attcaggcaa tatgttggta ctagctaacg gtgcgtgaaa ttggntttga ggtgtccaag 240
naaacttgca tatgacgagn accncngtag aacttgtatt agcctatagc actactgtct 300
acagtgtcca accaacntnt gattgaaaac aactacatta agaaccttag cctcgcgccc 360
agaatagnga gacctctgca tcagtgatta ccctctatac ctgtactctc tgagagagca 420
ccaaacgagc atgacagcga tatcnggaag agcccaatta ttcccagtga acagcgcgag 480
cgaccccggg atgaagcaga scaccccatt tccaatctgg ttccgtgcac cccccgcaat 540
agatccccgg cggtggaccc ggcccctcct tccctgcaaa cctcctccga gggccggccg 600
gccttcgcag ctacgctagt tgagtcggca tcgggatccg caaaaccacc aacgaatcct 660
gccggcagaa acgtgcgcga tcgcgtgacg gcggcacagc ctgcgactgc aagtctgaaa 720
tcggtggggt gggtacctag tggcgatggc gagagctagc ccaccacccg cgccggcaag 780
cggtagcagg ctccagttgt tgtactgtac ccacagcaac gggcaactcc accgaaccgc 840
acgcgaaagc aggagatcgg tgggggtgga gcgggggtgg ggagggtggg ttggattcga 900
agcttgcgtt gcacgggaca gatctacagc tgagcggtgg cgtgcgggca cagattggaa 960
tcgcccgccg acccagagcg cggggangga ggtgggcgaa t 1001
<210> 65
<211> 552
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (75)..(75)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (79)..(79)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (121)..(122)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (502)..(504)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (506)..(506)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (508)..(508)
<223> n представляет собой a, c, g или t
<400> 65
tagggtcctg ctacaagaga tcgccacatt ttattgctac ggaagtccag ttgtgtctgt 60
ctgtttggtg gtcantggna tatggttcgg tttttactgc tgtaaaaagg gactsgggaa 120
nnaaaaatgc aaactgactt ggattttttg ttctgttctg catgaagatg aaatggtagg 180
gtcgtcggag gaggacgaag catgctcggg aggagacacg gaggcgacgg agccggggca 240
gcaggagcac agctcccgcc tggcggaccr tgagctgaag gagatgctgc tgaagaagta 300
yagcgggtgc ctgagccggc tgcggtccga gttcctgaag aagaggaaga aagggaagct 360
gcccaaggac gcgcggtcgg cgctcatgga ctggtggaac acgcactacc gctggccgta 420
ccctacggta accatgcatg catcctggca aacacgcagc agcagcatcg ctcgctggaa 480
tgrcagatct gtgaccagca tnnncngncg gtgcaggagg aggacaaggt gaggctggcg 540
gcggcgactg gg 552
<210> 66
<211> 7437
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (4454)..(4454)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (4463)..(4463)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (4474)..(4474)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (4497)..(4498)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (4536)..(4536)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (4549)..(4601)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (5112)..(5112)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (5194)..(5194)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (5297)..(5299)
<223> n представляет собой a, c, g или t
<400> 66
aatgtcaagt atatccattt aaatatcatt aggtcccgtt tgtttccttt cattttaagg 60
aattggaatc ttactaataa aataagctat ttttttagaa tacgagattc caccactttc 120
caaagttatc agataagcct atctcaaatt catggggtga gagatggaaa ttgattctat 180
agatttacat gttattttcc cgatgtacaa cttatatcat actctcctaa ttgcttcgct 240
ataacataaa tgcactatat aactatctct cttatatgat ttaggataat atacaaatat 300
attacatata taaatatatt aacttaatta gttttgtcta aattataatt attaaaatgg 360
aattcaattc caacgaaaca aacgggccct tacaaaattt ctagtatcat ttaaccatct 420
attcaacaca ccaaagataa ttggataaaa tagcaacact aggacaaata ctacatagca 480
cattacatgt tccattatat ggtcattaaa ttgtcgttaa gccttctata acattatggc 540
ctaaaaggtt gttaaatagt aaaaaaagat ggatgactaa agtccaagtt catgcttggg 600
ctaagtttat attgggtatt ttataccacg ggttaacggg tatgggtgaa ggcggaacgt 660
tctgattccc gtttacttat tgggtgaaga tttttgccta ataatagacc tacgggtgaa 720
tatttatccc acatatatat cctagtggag tcaatatcca tcggatatcg gtcgtgggta 780
cccattgcca tctctagatc gaagagtaga aatttacttc ctaaacctct ctctgtctca 840
tgcagcacaa tagacgcttt gtttcgttgc aacagcttgt ttctctttga cgtccaaatt 900
cgtctatcta cggacccacg gccgcccaga ttttgaaatt tcaaaacgga acacaccccg 960
gggttcggag tatctgctgc ccctgcggtc ctggaaagcc cggcccacct ctcacttgca 1020
ccaccagctc accggttccg gtcaacagtc tctcgcgggt gcctgtgccg gtggtcctcc 1080
gtcctggctc tggctgccac ccacctcccg attaccgttc tcgcctcgac ttcaaaacaa 1140
gagcccagat ctaaaccaag cacgcccatc tttgccacac cacaccccca gtattcgaat 1200
ctctcgtgcc cagatgcggc aaattaaaaa caacggacag acgcggaacc cctccggcca 1260
acggatctca ccctctgcgg catgggtccc actcacgctc gggtccactc gacagcgtgt 1320
agggcagaga ggcgagcggt accagtacca taggcctccc gacgcagtcc gggcagcggg 1380
ccccgcggat tggaccggtc aaaaggcgtg gcccaaccaa accccaaggg atccggcgct 1440
ttgtctgcac gtgaatggtg ccaagatcgc ctggttgaca ggtgggaccc gtgaggttgt 1500
agacccacat gtctgtggcg ttaaaggagg ggggagggat cggcgggcgg gtggtgcgcg 1560
cgggcaggcg ggcgtcgcgt ggtggtggtg gtggtgggtg ctttgactgc aggcctcggc 1620
agcaggcaga gaggactaga ggagtcgggg cctcggagga ggggagggag agggcgaaga 1680
gtagggggaa ccaaatcttg aagggtaaac ggagagttct ttcgtggagg aggaaggggg 1740
ggacagcagg aggagggtag aggtatgtgc gcacccatct gttcttgctc ctgatttggc 1800
tgtttgtttt ttctgtctgt tcttcgctgt tggtagtttg tgaccgtgaa tgggcgttcc 1860
tggtccatgt tcgcgtgcgc tgctgccgat tctgggagct ctctggtcgt ccgtctcgct 1920
gggatctgcc ttttccccgg tgagagccgc ggaacgttcg ccgccttttc ttactcgcgg 1980
gccagttatg gtttctggag cgttttctct gttcttggcg aggtggtcat cgctctgaga 2040
acgatgcgct ctttctccga gtttgtgctc aagttttcgt cagcctagag gctatagcgt 2100
ttgctgcgga tctcacgact tctctcttcc tcttctctat tggtgcatac gttttcatcc 2160
gaaatccatt agttagtgcc cgagccgtca attctttgtg gatttgcttg ttccccttcg 2220
ttacaggctc ggaaatgccc ctgaacagat tcacaggggt cctagattag gattattttc 2280
tatgactttc caagagtcag gagcacgatt gctttctctc ggctgtctgc ctggttcatg 2340
actcagccgg gtttgcaagc ctaggaagaa cttgctcacg tttcttacat ttatctagat 2400
tcgagggacg ggttgtactc gttaacaaag ttcacctcgt tagtcattaa agctccgctg 2460
ttgtgaatga tgctgccatt gcgatatctg gaatcatcgc tctgatcgat ttggttgtta 2520
atccacttac aggtagctca atagatctac tgctctcggg ggagttaatg caaagctgag 2580
ttgctgcacg ttggctttct tcagagatgg cttcagctgg tgtagcccca tctgggtaca 2640
aaaacagcag cagcactagc attggtgccg agaagttgca agatcagatg aacgagctaa 2700
agattagaga tgataaggtg aagatgcctt gatatcttgt ttcgggctta ctgtaatttc 2760
ctcaagatta tgtgaaaaat gggactgtga tgtaaccttt ggtgtgaatg ccaaatgcag 2820
gaagttgaag caaccataat taatgggaaa gggactgaaa ctgggcacat aattgtcacc 2880
actactggtg gcaagaatgg tcaaccaaaa caggtgagtg ctttactgca tttgatcatg 2940
atttatcaac tattctacat gtttttagtg catgtctgaa tctaataatt gagagtcaag 3000
accataattt aatgtccttc ttttgcatat tgccaatata tccatgttgc taacttataa 3060
gattgtggag ttgttctgat cagttttgtc agattctttt tgtataataa tgtgtattta 3120
ttggttgcat ttgcagacag tgagctacat ggctgagcgc attgtaggtc aaggttcttt 3180
tgggatcgtc ttccaggtta tttgcaataa cttgtgactg actttgatat gtactattat 3240
gtagccgcct gtggtgttgc tttccacggc gctgcacatg ttttagatct tcatatcttg 3300
cgtgctataa atcacctttc ttaatcagat gccatttcac ctgttcatag gctaagtgtt 3360
tggagacggg tgagactgtt gccataaaga aggttcttca ggataagcgt tacaagaacc 3420
gcgagttgca gaccatgcgc cttcttgacc accctaatgt tgttgctttg aagcattgct 3480
tcttttcaac taccgagaag gatgagcttt atctgaactt ggtccttgag tatgtgccgg 3540
agacagttca tcgagttgtg aagcatcaca acaagatgaa ccaacgcatg ccacttattt 3600
atgtgaagct gtatatgtac caggtaatgg tttgtcctgt tcctttttgc tgttgtttta 3660
attatacctt aaagcttatg ttttgggccc tgtttgatgt tgaaactaac aaacatattt 3720
catttcgcct aaatattgtc tgctccaatg aatgtgctag ttctttttca atatttgata 3780
ttatattgga ttttggcaga tatgtagggc attggcttac attcatggca ctattggtgt 3840
ctgccacaga gatattaagc cacaaaacct tctggtatgc tggaaaatct gctattttgc 3900
tactgtatct ttttgtaaag aaatgatttg tactttgaaa ttgatgttca aacttcacta 3960
caggtgaacc cacacaccca ccagcttaaa ctatgtgact ttggcagtgc aaaagttctg 4020
gtcaaggggg aaccaaacat atcgtacatc tgctcccgat actatagggc tccagagctc 4080
atatttggtg ccactgagta taccacagcg attgacattt ggtctgctgg atgtgttctt 4140
gctgagctta tgctagggca ggtaaggtgt ctcaaatttt tattgccatt ttaaaaaagg 4200
ttttcaagcc aacaaggtcc tttcagttca cactgtctta caagaactat ttggacagcc 4260
tttgtttccg ggtgaaagtg gtgtggacca acttgttgaa atcatcaagg taattgtcgg 4320
ttctacaagc ttgtgaattg tcttctatag aagcataaaa tctgatcacc cctaaaatga 4380
ttttgtatgg caggtcctcg gtacgccaac aagggaagaa attaaatgca tgaacccaaa 4440
ttacacagag tttnaagttc ccnacaaatc aaangcacac ccatggcaca aggtgcnnaa 4500
atctktctac attttgttac aatactctaa gaaaanctgt tactgttgnn nnnnnnnnnn 4560
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn ntgttactwa tttacttttt 4620
gtacatttta tctttcaggt mttccacaaa aggatgccgc cagaagctgt tgatctggtc 4680
tctcggctac tccagtactc cccaaatctg agatgcactg ctgtaagtgc atgccattgt 4740
acattataca tgatggaaat acccctgttg actttggttt tctaagatct tyatgaatgt 4800
tttgtccaga tggaggcact tgttcaccca ttcttygatg agcytcgaga tcctaatact 4860
cgccttccaa atggtcgctt tttgccacca ctattcaatt tcaagcctca cggtatgttt 4920
catgcctaca taattcaaca tcgttatcat agctgctaca accaggtakc agtgtagtwc 4980
yaagtttgtt ctttgtatat caccacctta catgctcgcc acctctgttc tgcagaactt 5040
aaaggagtcc catcagacat tgtcgcgaaa ttgattccag aacatgcaaa gaagcaatgc 5100
tcctatgttg gnattgtgaa atgaccgcgc cttgagactg gaacctgtgg ttgcaattgt 5160
gaatttcccc tgggatgttt gacgatctga ggcnatgcga gcctgttgtt gaagatgcaa 5220
ggttacgtac ttgtacgaca atgtgacctg tgtagctgag tagtctatgt cgcagtgaca 5280
tgtaacggca ccccccnnnt tcctactaac tgacgcttac tcgagattgc catagttgat 5340
cttgtaattt gttatagagc agtatgaatg tatttatggt agcttgaatc tatgtatgga 5400
ttcacttcgt ttttccatgt ttccttgtct ccagacccag attgctaccg tattgtttca 5460
gaattcctag ctacctgttg cctattgagt attgactacc agcttgcact tgtctgttat 5520
tgcactggct gtggaatcag ctgttgattt ttgccacaat attttagttc agatgtactc 5580
cctattctaa aaagaatgtg aaatcttact aatagaatag actacttttt ttagaatttc 5640
tttccatttt gaggaattaa aatcttacta atagaataga ctactttttt ttagaatgtg 5700
acattacacc actttctaaa gttatcatat aagcctatct catttatggg gtgagagatg 5760
aaaattgatt atatagattt acatactgtt tttccgatgt acaatttata gcacaccctt 5820
ctacttgctt cgctataaca taaatgtagt atataactat ctctttcatg tgatttaaga 5880
taatatataa atatattaca tatataaata tatgaactta attagtttta tctaaattat 5940
aactattaaa ataaaattca atttcaacga aacaaacggg gccttgatta attataaaat 6000
gtatttttgt aataagttga tttaaagcta taatgtaaat actatttact agaaacttgg 6060
ttaaatatga attagtttaa ctaacgagtt taattggcat accacttata gttatattct 6120
ttgagacgga gggacgagta cgttgttcga tcggtctgga agtatgctga cttgatcgtt 6180
cttaccagaa agttgcatta ttgcagcgtt tgagacgact gacgaggaaa tgtgacacgc 6240
agatgctact cagtgcttgg caggactgca ttccaagtgg tccttctggg gagagaggaa 6300
tcatagactg tagctccggt ttcttgaaaa aaaacggttc ccgtgaaatg gcaggtatgg 6360
ttctccggtt cctttgaaaa ctactctttg taaaatgaag tatgcttggc tctatcgaag 6420
ttagctgttg ttaacagcca taccagacag gttctttcag tgtccggtta gattttgagg 6480
cgtcgagggt tgtttggttg agaagtggag agttccttta gagtgtgttt agttgagaag 6540
tggaggaaaa tggatcgact atattcctat tttttttatg tttagtttcc aagaaaagcg 6600
gagcagagcg gctcctgaag ttttagaaat ttaccataaa tagtttaaat gctcccgctc 6660
cgtcaaaacg aacatacacg agcgctctcc tccctctact tccttctaca accgtatgtc 6720
tttccaatca agcaaagaac ggagtagctc tgctctattc tactcttaac caaacaaaaa 6780
aatgaagtga ctctgttctg cttgtcaaat gcgaaataga atgattctat tctaaaaaat 6840
tggaatagag ccgctccaac caaactaacc tcactcgagg gactaaagtt tagtctttac 6900
tctatttgat tccaaggact aaaagtattc ataacatatt aaatgacttg aaaactaaaa 6960
tgttcttaac attcttccgc cattagcata actaaaataa actagggata agtgaaatta 7020
atatggacta aaacaatttg gtcgctgttt tattcccata tttgacaatt tagaaattaa 7080
ataaaactaa aatagatgga ttaattttta gttcctcaaa caattttttc taacaatttt 7140
cgatggacta agtttagtca tttttcataa gaaattaata tggactaagg gcctgtttgt 7200
ttacccctca gattatataa tctggattaa ataatcctaa gaggcaaaca aacagtctag 7260
cttatttgtc gagattatat aatctaactc ctggattatg ataatccata agcaagtgag 7320
gaggtgctta tttcagatta tttttttcca cttctccact accctttcaa gtttcctaga 7380
aattacccac cattgccatt ataacccacc attggcattc ttgtcttcct catacaa 7437
<210> 67
<211> 570
<212> ДНК
<213> Zea mays
<400> 67
aaggtgggaa aactgaaaaa gacaatcatg aaggtgtaaa ttctttacta tctgaggagt 60
tggaaaaact agctaatggg aatagcaagg tatgatatac cctccatgat tctgctttca 120
tttattcttt gttcatatgg tatggttatt taacagtgct atgtattgcc gtaaatgcag 180
attcctggta cattagatga gtatagaaag cttgtcrttc caataattga ggagtatttt 240
agtacaggag atgtggaatt ggcagcttct gagctgaagt gtcttggatc tgatcagttt 300
catcattact ttgtgaagaa gcttatatct atggcaatgg atcgccatga caaagaaaaa 360
gaaatggcat cgattctgtt atcatctttr tatgctgatc tactgagctc ctacaggatc 420
agtgaaggtt ttatgatgct tctggagtct acagaagatc taactgttga tataccrgat 480
gctactgatg tattggcagt ttttattgca cgggctattg ttgatgaaat tttgcctcct 540
gttttcctca ctcgagctag ggcactactt 570
<210> 68
<211> 904
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (87)..(89)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (94)..(97)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (138)..(138)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (145)..(145)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (257)..(258)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (263)..(263)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (319)..(319)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (424)..(424)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (441)..(444)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (448)..(448)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (451)..(459)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (461)..(461)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (464)..(465)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (494)..(494)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (547)..(547)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (552)..(552)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (557)..(557)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (602)..(607)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (634)..(637)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (647)..(647)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (652)..(654)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (657)..(657)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (659)..(659)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (665)..(665)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (682)..(682)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (691)..(691)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (693)..(693)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (746)..(746)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (806)..(806)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (842)..(842)
<223> n представляет собой a, c, g или t
<400> 68
atgcgccctt gggtacatag caggagactg gaaaggtaac aaatggaata cggacacgta 60
gataaccatg gaggcccagc aatgttnnng aggnnnntat tgtttggtca ctgcatagcc 120
gatgatgatc accacgcnct cgtgnatgcc tcatgtctta acagcagcca gatgatctct 180
grgcccggtt gccacctacc acatccacgg tatctggcac ataccttcaa gaacaaatcg 240
ataaggtcaa aaaaaanngg ggnacggctt ttacatagat aataaaggac cagcacaggg 300
aaacaaatra aggaaatcna aaagtgattr atgattttac atagatataa cactgaaaac 360
gagaccagca gaagaagcta gtcttattgc agcagctaat gatgccaacc ctggtacacc 420
cccnagaaag aggatcaaca nnnngaanag nnnnnnnnnt ngsnncttat tttgctgatg 480
actaacaact ggangcaaaa agaccaacag aaggaccggt tgatattaaa aatgtaatta 540
cttttancag cnagcgnctc cggttttcta gtacttgcag caactaatgt cacataaaaa 600
cnnnnnntgt caaatgccaa tcaaacacac aaannnngaa acgaganatc annnagngnt 660
gtgcnttaca attcttcctc cnggttcatt ncnttcagag cattctttac aatacactgg 720
aaggcatctt ccacgttcgt tccatncctt agcagacgtc tcaaagtacg ggatgttccc 780
tttagaggcg caccatgcct ttgccntttt tctccgacac ctaaaaccaa agcagattat 840
antttctaag cagctcgtga tccagttcaa gagaagaatt tattcagaga acaaatcatg 900
tagc 904
<210> 69
<211> 426
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (310)..(310)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (316)..(324)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (348)..(348)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (396)..(396)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (405)..(407)
<223> n представляет собой a, c, g или t
<400> 69
tcttcagtct ccaccctgat tcaacaacag actctgacag cttgcacggt agctgccccg 60
ycatcgaagg ccagaagtgg tccgcgacta aatggatcca tgtgaggtca tttgacctca 120
ccgtcaagca gccgggtccc tctgatggat gtgaggacga caatgtcctc tgcccccagt 180
gggcggccgt gggcgagtgy gccaagaacc ctaactacat ggtggggacc aaggaagcac 240
ctggcttctg ccggaagagc tgcaaagtat gcgcagagta aggtatcggt cctctgcgtc 300
tgatgagtan tcgtgnnnnn nnnnttacgt agttgctgtc accatttnac cagggtttag 360
atacgaccga gtacagcatg tataagacag tacaancccg gaagnnngag tcgtaagagt 420
tagggg 426
<210> 70
<211> 595
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (201)..(204)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (458)..(461)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (569)..(576)
<223> n представляет собой a, c, g или t
<400> 70
cgtccgcggc agcggccgcg tccaggtggt cgggcccgac ggggtgcgcg tcctggagac 60
gcgsgtcgag ggcggcttcc tcttcatcgt gccccgcttc cacgtcgtct ccaagatcgc 120
cgacgcgtcc ggcatggagt ggttctccat catcaccacc cccaagtaat ttgttgtctc 180
gatcgatcga tccatcgatc nnnntttttt tattgcgaat tgcactggag atttgattgc 240
acgtgaatta atgyttgcat tgcattgcag cccgatcttc agccacctgg ccgggaagac 300
gtcggtgtgg aaggccatct cggcggaggt gctgcaggcg tcgttcaaca ccacgccgga 360
gatggagaag ctgttccggt ccaagaggct cgactcggag atcttcttcg ctcccccatc 420
caactgagaa aataggccgg aagccccacg gtggagtnnn ncctctcgtt aggtcgtcgt 480
gcttagatta ggttagctag cttgccttta ataaaaagag agtggtggtc gtcggcgtcg 540
gcttcggcgg tctgcttctt cttcattcnn nnnnnnagtg cgtcggtcgg tttag 595
<210> 71
<211> 558
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (39)..(39)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (58)..(58)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (60)..(60)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (67)..(67)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (80)..(80)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (82)..(82)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (97)..(97)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (104)..(104)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (111)..(111)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (116)..(116)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (138)..(138)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (150)..(150)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (155)..(155)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (157)..(157)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (159)..(159)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (166)..(166)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (172)..(172)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (174)..(174)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (186)..(186)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (191)..(191)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (193)..(193)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (196)..(196)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (205)..(205)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (208)..(208)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (212)..(212)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (215)..(215)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (217)..(217)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (219)..(219)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (228)..(228)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (231)..(231)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (233)..(233)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (247)..(247)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (255)..(255)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (258)..(258)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (261)..(261)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (265)..(265)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (278)..(278)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (280)..(280)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (292)..(292)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (321)..(321)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (323)..(323)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (329)..(329)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (331)..(331)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (335)..(335)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (348)..(348)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (370)..(370)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (393)..(393)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (395)..(395)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (403)..(403)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (413)..(413)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (432)..(432)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (447)..(447)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (467)..(467)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (472)..(472)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (474)..(474)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (477)..(477)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (494)..(494)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (505)..(505)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (508)..(508)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (526)..(526)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (529)..(529)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (550)..(550)
<223> n представляет собой a, c, g или t
<400> 71
ccgtgttggt tggatctatg gctcggtgac ggaggatgnt ggtcactggg taccggantn 60
gcacaanccg gggttggaan gntcggtgta ctgtgtnyac caangcgtga ncgccnttcc 120
gcggcaccgc gcccatcnaa cctgacygan ccgtncntnc caccanggtg cntnccggtg 180
ggctanctgg nantcnagtg gagantcntt cnttncntnc ccgmaacnaa ncngcgctgc 240
tggcgangcc gcagnaantg naagnttctt gcagaggnan tcgcgtacct gnaacgtggg 300
tatctacccg ttcacgtcca ntncttccnt ngatncgtct actgcttncc tgccggcgct 360
gtcgctgttn ctcggggcag ttcatcgtga agnancgctg aancgtgacg ttncctgacg 420
tacctgctgg tngatcacgc tgacgcntgt gcctgctggc ggtgctngga gnantcnaag 480
tggtcgggga tcangyctgg aggangtngg tggcggaacg agcagnttnc tggctgatcg 540
gcggcacgan gcgcgcac 558
<210> 72
<211> 1193
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (8)..(8)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (15)..(15)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (25)..(25)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (79)..(86)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (88)..(88)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (100)..(102)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (124)..(126)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (135)..(135)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (239)..(242)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (357)..(357)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (365)..(365)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (379)..(379)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (470)..(470)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (496)..(503)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (506)..(507)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (512)..(513)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (519)..(524)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (571)..(579)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (623)..(625)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (670)..(670)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (691)..(691)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (806)..(808)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (841)..(841)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (847)..(848)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (850)..(850)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (968)..(975)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (1016)..(1018)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (1083)..(1083)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (1108)..(1108)
<223> n представляет собой a, c, g или t
<400> 72
gtgaatgnac agggngactc ctggnacagc ctactcggca ggaacaagca cgacgaccag 60
gagaagaaga accagcagnn nnnnnngncg gaggaggagn nnctggcgac cggcatggag 120
aagnnngtca cggtnggccg agcccgacca caaggaggag ggacacgagg ccgccgagaa 180
gaaggacagc cttctcgcca agctgcaccg caccagctcc agttccagct cggtgagtnn 240
nntcgtcgta aaacatgayc tgctgctagc tagtttaatt gactccgcct tcggawcagt 300
aagctaataa accggcttct cactgcgatc gtggtgcctg cgcgcatgca gtcgagncga 360
cgacnaggaa gaggaggtng atcgatgaga acggcgarat tgtcaagagg aagaagaaga 420
agaagggcct taaggagaag gtcaaggaga agctggcggc ccacaaggcn ccacgatgag 480
ggcgaccacc accagnnnnn nnnacnngcc cnngcgccnn nnnngcccgt ggtggtggac 540
acgcatgctc accaccagga gggagagcac nnnnnnnnnt tcccggcgcc ggcgcctccc 600
ccgcacgtgg agacgcacca ccnnnccgtc gtcgtccaca agatcgagga cgacgacacg 660
aagattcagn accccaccac aggcaccgga ngaggagaag aaaggcctgc tggacaagat 720
caaggagaag cttcccggtg gccacaagaa gccggaagac gctgctgccg ccgccgccgc 780
gccggccgtc cacgcgccac cgccgnnngc gccgcacgcc gaggtcgacg tcagcagccc 840
ncgatgnngn caagaagggc ttgctgggca agatcatgga caagataccc cgctaccaca 900
agagctcggg tgaagaagac cgcaaggacg ccgccggcga gcacaagacc agctcctaag 960
gtcgcagnnn nnnnncgtgt gcgtgtccgt cgtacgttct ggccggccgg gccttnnngg 1020
gcgcgcgatc agaagcgttg cgttggcgtg tgtgtgsttc tggtttgctt taattttacc 1080
aangtttgtt tcaaggtgga tcgcgtgngt caaggtccgt gtgctttaaa gacccaccgg 1140
cactggcagt gagtgttgct gcttgtgtag gctttggtac gtatgggctt tat 1193
<210> 73
<211> 774
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (281)..(283)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (328)..(328)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (652)..(652)
<223> n представляет собой a, c, g или t
<400> 73
agcatcatgg agtacggtca gcaggggcag cgcggccacg gcgccacggg ccatgtcgac 60
cagtacggca acccagtcgg cggcgtcgag cacggcaccg gcggcatgag gcacggcacg 120
ggaaccaccg gcggcatggg ccagctgggt gagcacggcg gcgctggcat gggtggcggg 180
cagttccagc ctgcgaggga ggagcacaag accggcggca tcctgcatcg ctccggcagc 240
tccagctcca gctcggtaat tacgactctg gatacttctt nnntcttttg tgtgcgcgct 300
gcttcgtcct atatataata atacatgnag ttaggcttag taataatcaa ttaatttaat 360
ccgtgggttt cgtgtttaag tcggaggacg acggcatggg cggaaggagg aagaagggaa 420
tcaaggagaa gatcaaagag aagctgcccg gaggccacaa ggacgaccag cacgccacgg 480
cgacgaccgg cggcgcctay gggcagcagg gacacaccgg cagcgcctac gggcagcagg 540
gacacaccgg cggcgcctac gccaccgrca ccgagggcac cggcgagaag aaaggcatta 600
tggacaagat caaggagaag ctgcccggac agcactgagc ggcgcctata cntggctgtg 660
ctgtgctgtg ctggcgcgtc aaagccgtac tcttcagygt tccatagata ataagataaa 720
cccatgaata agtgtcccta ccctttgatc atgtgacagg gacagggaca ggga 774
<210> 74
<211> 885
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (140)..(141)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (211)..(212)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (662)..(663)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (675)..(675)
<223> n представляет собой a, c, g или t
<400> 74
taggtattgt acacgctcta gcttgacaaa tggtcagccg ttgatctctg ctatttgcaa 60
ccagagctta agttcatctt ggattcatgc aggtcctggt gctccaacca tattcaactg 120
cgtaaggcca ccctgtcatn ngcttgactg gtcctcttgt gatatgttca tgttaatagc 180
atratgtctt ttgttctatt ggaaaataaa nngtctccct ggactctaaa atcaatgcct 240
gtgaacacat gaactgtttg tgtcacccat gttcctctgc tccttggcac tttctgatgc 300
atgctcaaat gcttaagaaa gactcataga agcgactcct attcctatgc caggtcattg 360
agataccaag gggcagcaag gttaaatatg aacttgacaa gaaaactgga ctgatcaagg 420
taaagcaatg ttgttttcct cccgctgaag tcttattgtg aagctatatt tcttgccagt 480
tctaatattt actcctttcc gtttcaatct gtgtgcatgt gcaggtggac cgtgtgctgt 540
attcatcagt tgtttaccct cacaactatg gattcattcc tcgcacgctt tgtgaagaca 600
gtgatccttt ggatgtactg gttataatgc aggtatgctt cttttttata tatatcattg 660
gnngattcac aaaantggta catcagtagt gatctgagta tccttgggca taagttgagc 720
taattttcaa atcttgtcat tttccatttc tgsgaatggt cgagaacatg tctataaact 780
gttacttcca agcatgtagg agccagtcat tttccatttc tgtttatagt tgcctagtcg 840
ggaacatgta tgtaaactgt tacttccgtg catgcaggag cctgt 885
<210> 75
<211> 935
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (84)..(84)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (219)..(221)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (303)..(305)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (783)..(783)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (804)..(804)
<223> n представляет собой a, c, g или t
<400> 75
atcaagagca gcagctgctt tgctgagaaa caggctgacc ccgcatttgc acagttgcag 60
gcctactagc accacttgca gctnggttga cttgccatcc tatccaacaa ggagaatgaa 120
gaatgattag gtgctcccgt atacagataa acaatagcaa acatastgat catgggattc 180
atggcttatt tttcactttg aatcatatgc aatattatnn ngtwgcacag tgttctttgt 240
ttgtactcag tccctcaata aaagagggcc tccatatgtt gacatactat acttgatgac 300
tcnnnaagga atgagaaaat gctgccacaa aaaagtctac aacacaaatg atctagttac 360
ctgttcttta tctcccctgc catggtcatg aatgccttct ccacatttgt tgcatccttg 420
gcactagtct caaggaatgg tattccgatg tcatcagcaa gggcctatga tgacaagcaa 480
catgcagcca atttaactat catcccggtt gaaagaagca tgtccagtaa aagtaattaa 540
tgcagagaaa tattaccttg ccagcctcgt aagaaactac tctgttctca gccaggtcac 600
acttgttccc caccaaaagc ttgttcacat tttcactggc atacctatcr atttcattca 660
gccactgctt gacattgtta aagctctcct ggtcagttac atcatacaca acctatagaa 720
atacaaaagt ttaaacaaga ctcagattaa caaagatgag ataatagcag ataggaaaaa 780
aancgaaata agaaaaagaa agcntcacaa taatgccatg agctccacgg tagtagctgc 840
ttgtgatggt cctaaagcgt tcttggccag cagtatccca ctaaatcaga araatgtgga 900
gaaacataag tgtcaaagct tctaactgtt aggaa 935
<210> 76
<211> 710
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (593)..(596)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (598)..(600)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (606)..(607)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (610)..(617)
<223> n представляет собой a, c, g или t
<400> 76
agcatsacca gaagcagagc ctgatggaca aggcgaaggg gttcgtygyg gagaagatcg 60
cgcacatccc caagcccgag gcsacgctgg acggcgtgac gttcaagggc ctgagccggg 120
agtgcatcac gctgcacagc agcgtgaacg tgtccaaccc ctacgaccac cgcctcccca 180
tctgcgaggt gacctacacg ctccggtgcg ccggcaagga ggtggcgtcc ggcaccatgc 240
cggaccccgg ctggatcgcc gccagcggct ccaccgcgct ggagatcccc gccaaggtgc 300
cctacgactt cctcgtctcc ctcgtcaggg acgtcggccg ggactgggac atcgactacg 360
agctccaggt cggrctcacc gtcgacctcc ccatcgtcgg caacttcacc atcccgctct 420
ccacctcygg cgagttcaag ctccccaccc tcaaggactt gttctgatct agtagtagct 480
cgcttgcctt stgttctgtg cgggcgcgca ccagcgatct gtacgacgas cttttgcaaa 540
taaamgamgc agctcctctg ttctatatat ctmagkgrat gsmtrrkyta aknnnntnnn 600
tgrytnnryn nnnnnnnaaa taaagagctg gatttcrttc aggttcctgt ctcyaagctg 660
gattycatts gggcatccac crtgatstgg atgtgcctgc cgcgtccgtc 710
<210> 77
<211> 663
<212> ДНК
<213> Zea mays
<220>
<221> другой_признак
<222> (24)..(24)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (101)..(101)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (159)..(159)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (229)..(229)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (249)..(249)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (252)..(252)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (264)..(264)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (267)..(267)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (294)..(294)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (439)..(439)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (462)..(462)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (588)..(588)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (594)..(594)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (616)..(618)
<223> n представляет собой a, c, g или t
<220>
<221> другой_признак
<222> (641)..(641)
<223> n представляет собой a, c, g или t
<400> 77
ttcctctata agtacccgcc ccanatctgc gccattttct catcgcagaa atcctccgca 60
cttcacagcg tatcatcgtt ttycatcgct cctactccta ncatccagaa aatctgagmg 120
gtattgatgg cgcccaaggc ggagaagaag ccggcggcna agaaggtggc ggaggaggag 180
ccctcggaga aggcggctcc ggcggagaag gcccccgcgg ggaagaagnc caaggcggag 240
aagcggctnc cngcgggcaa gtcngcnggc aaggagggcg gcgacaagaa gggnaggaag 300
aaggcgaaga agagcgtgga gacctacaag atctacatct tcaaggtcct gaagcaggtg 360
caccccgaca tcggcatctc ctccaaggcc atgtccatca tgaactcctt catcaacgac 420
atcttcgaga agctcgccnc ggaggccgcc aagctcgccc gntacaacaa gaagcccacc 480
atcacctccc gcgagatcca gacctccgtc cgcctcgtcc tccccggcga gctcgccaag 540
cacgccgtct cggagggtac caaggccgtc accaagttca cctcgtcnta gccnccttgt 600
wgtaggcgtc gttgtnnnct gcttctcaag caagcactgt natgtgccgc ttctcatggc 660
agt 663
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ВОССТАНОВЛЕНИЕ ФЕРТИЛЬНОСТИ У РАСТЕНИЙ | 2019 |
|
RU2831789C2 |
| КОМПОЗИЦИИ ДЛЯ ИНДУКЦИИ ГАПЛОИДИИ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2816649C1 |
| ГЕН-ВОССТАНОВИТЕЛЬ RF4 ДЛЯ ЦИТОПЛАЗМАТИЧЕСКОЙ МУЖСКОЙ СТЕРИЛЬНОСТИ (CMS) C-ТИПА КУКУРУЗЫ, МОЛЕКУЛЯРНЫЕ МАРКЕРЫ И ИХ ПРИМЕНЕНИЕ | 2011 |
|
RU2729107C2 |
| ГЕН-ВОССТАНОВИТЕЛЬ RF4 ДЛЯ ЦИТОПЛАЗМАТИЧЕСКОЙ МУЖСКОЙ СТЕРИЛЬНОСТИ (CMS) C-ТИПА КУКУРУЗЫ, МОЛЕКУЛЯРНЫЕ МАРКЕРЫ И ИХ ПРИМЕНЕНИЕ | 2011 |
|
RU2747979C2 |
| НЕДЕСТРУКТИВНАЯ ГЕННАЯ ТЕРАПИЯ ДЛЯ ЛЕЧЕНИЯ MMA | 2018 |
|
RU2820602C2 |
| ЛЕЧЕНИЕ И ИНГИБИРОВАНИЕ ВОСПАЛИТЕЛЬНЫХ ЗАБОЛЕВАНИЙ ЛЕГКИХ У ПАЦИЕНТОВ С АЛЛЕЛЯМИ РИСКА В ГЕНАХ, КОДИРУЮЩИХ IL33 И IL1RL1 | 2018 |
|
RU2776241C2 |
| НЕ ЯВЛЯЮЩИЕСЯ ЧЕЛОВЕКОМ ЖИВОТНЫЕ, СОДЕРЖАЩИЕ ГУМАНИЗИРОВАННЫЙ ЛОКУС TrkB | 2018 |
|
RU2800428C2 |
| ЦЕЛЕВАЯ ГЕННАЯ ИНТЕГРАЦИЯ ГЕНОВ-ИНГИБИТОРОВ NK ДЛЯ УЛУЧШЕННОЙ ИММУННОЙ КЛЕТОЧНОЙ ТЕРАПИИ | 2018 |
|
RU2797304C2 |
| КОМПОЗИЦИИ И СПОСОБЫ УЛУЧШЕНИЯ ТОЛЕРАНТНОСТИ К РАСТРЕСКИВАНИЮ СТРУЧКОВ У КАНОЛЫ | 2020 |
|
RU2834241C2 |
| СПОСОБЫ УЛУЧШЕНИЯ ЗДОРОВЬЯ РАСТЕНИЯ С ПРИМЕНЕНИЕМ СВОБОДНЫХ ФЕРМЕНТОВ И МИКРООРГАНИЗМОВ, ЭКСПРЕССИРУЮЩИХ ФЕРМЕНТЫ НА ПОВЫШЕННОМ УРОВНЕ | 2017 |
|
RU2802848C2 |
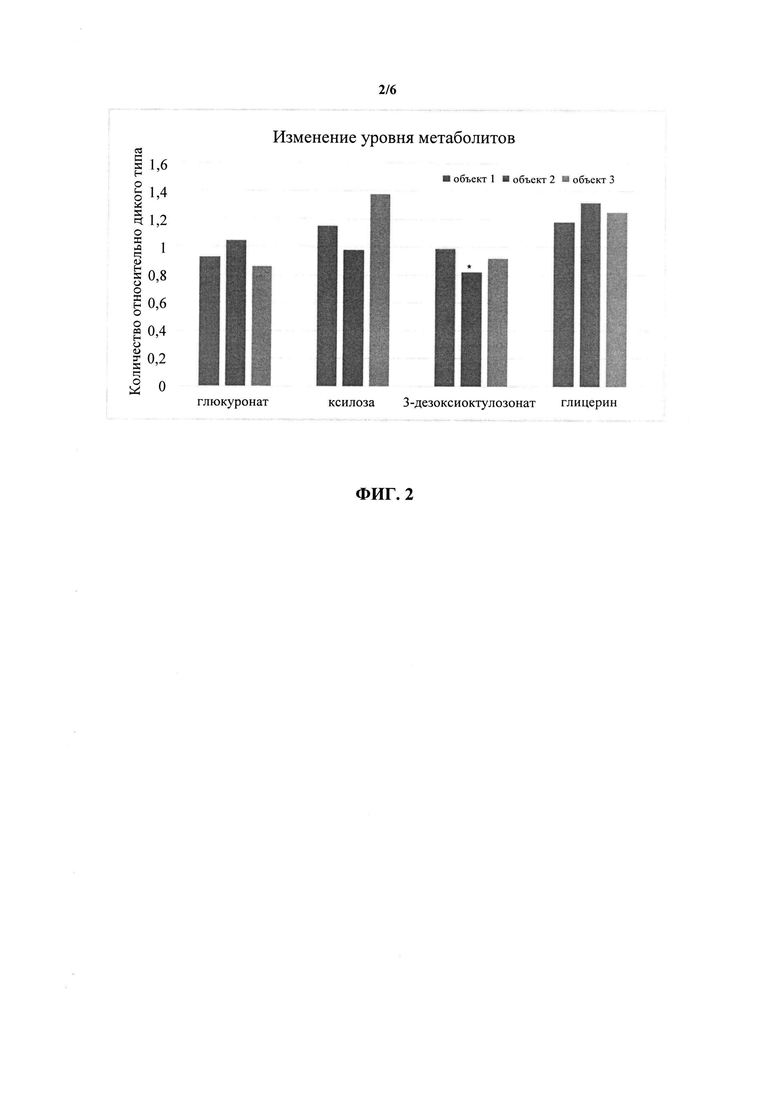
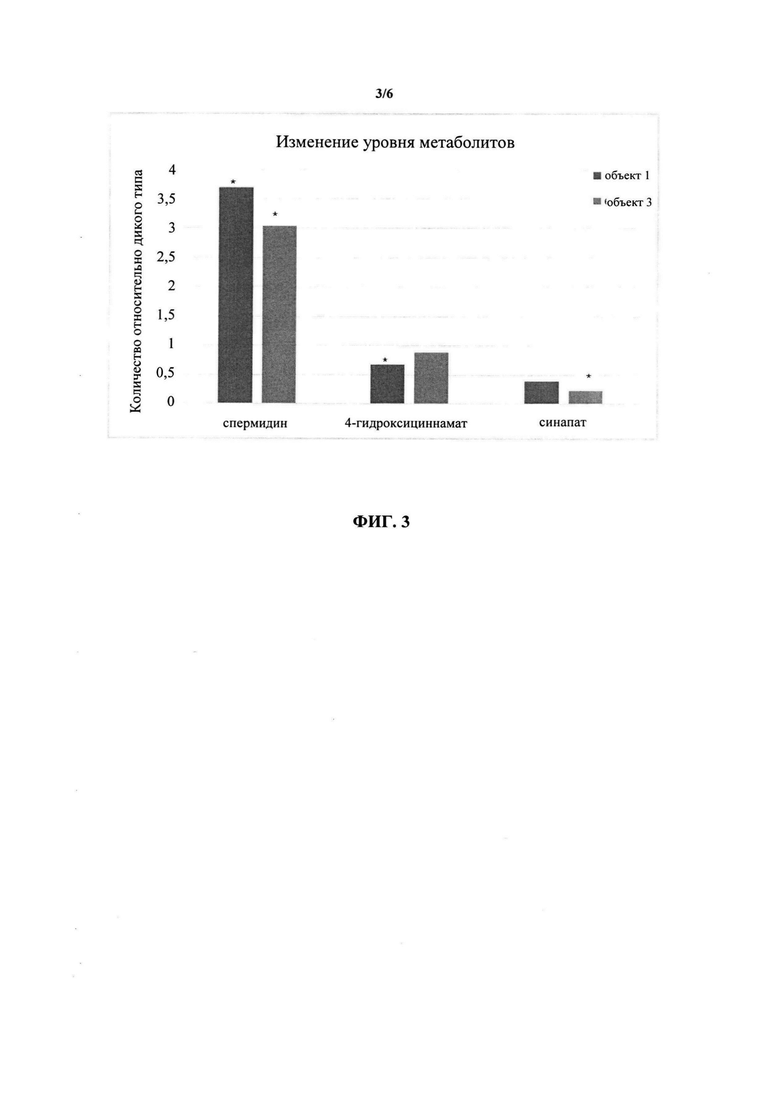
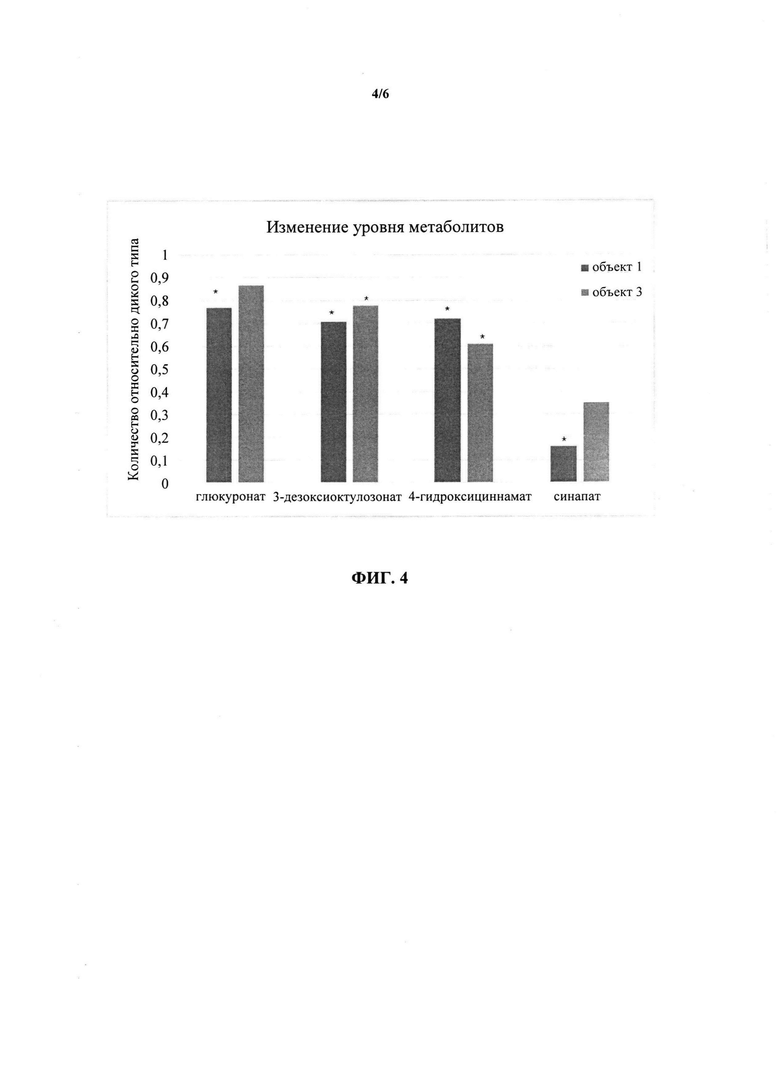
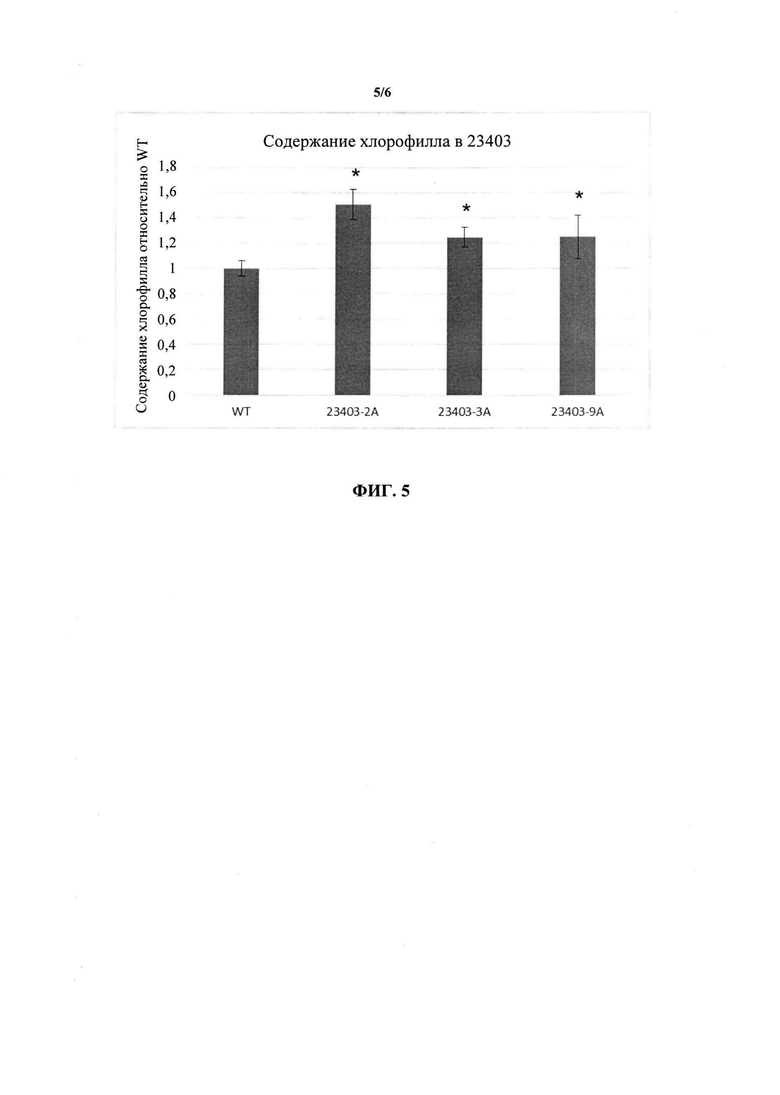
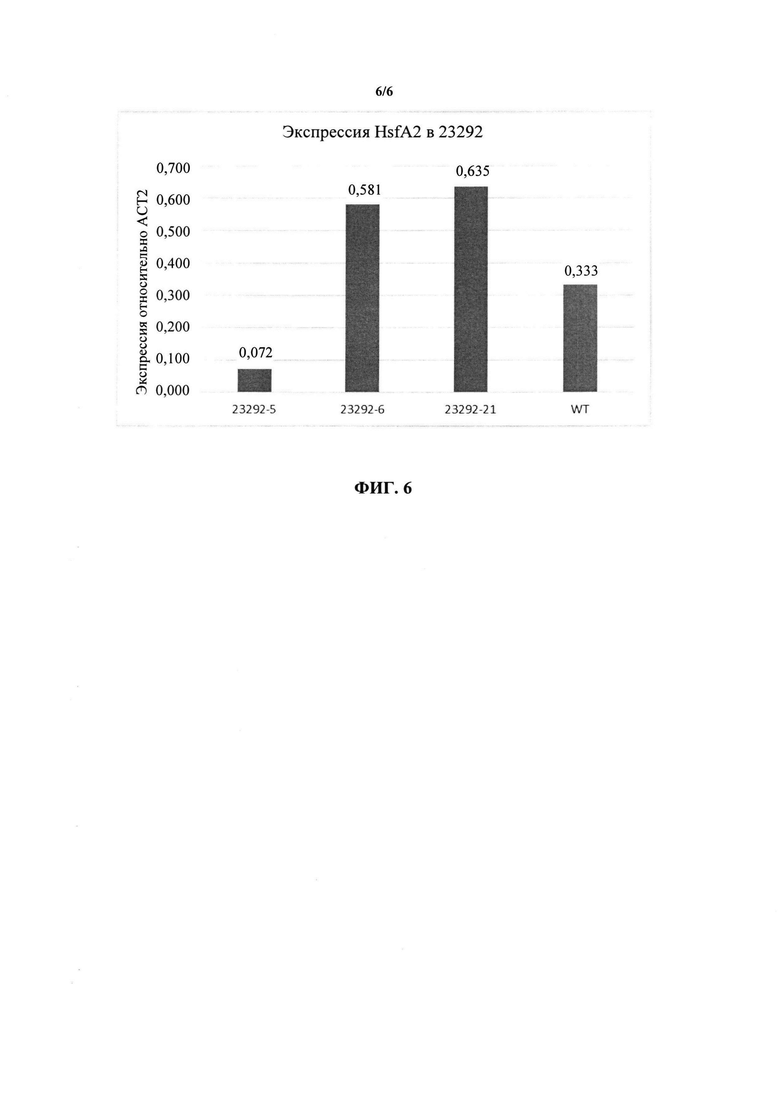
Изобретение относится к области биохимии, в частности к способу идентификации растения маиса, характеризующегося повышенной урожайностью. Изобретение позволяет эффективно увеличить урожайность растения маиса. 7 з.п. ф-лы, 12 табл., 6 ил., 3 пр.
1. Способ идентификации растения маиса, характеризующегося повышенными урожайностью в условиях засухи и/или урожайностью, которая представляет собой увеличение количества бушелей на акр по сравнению с контрольным растением, причем способ предусматривает стадии:
a) выделения нуклеиновой кислоты из первого растения маиса;
b) выявления в нуклеиновой кислоте из a) по меньшей мере одного молекулярного маркера, который ассоциирован с повышенной урожайностью в условиях засухи или повышенной урожайностью в условиях, отличных от засухи, где указанный аллель локализован в пределах 20 cM, 15 cM, 10 cM, 9 cM, 8 cM, 7 cM, 6 cM, 5 cM, 4 cM, 3 cM, 2 cM, 1 cM или генетически сцеплен с аллелем урожайности, который
соответствует любому из:
(i) SM2987, расположенного на хромосоме 1 маиса, который соответствует аллелю G в положении 272937870;
(ii) SM2991, расположенного на хромосоме 2 маиса, который соответствует аллелю G в положении 12023706;
(iii) SM2995, расположенного на хромосоме 3 маиса, который соответствует аллелю A в положении 225037602;
(iv) SM2996, расположенного на хромосоме 3 маиса, который соответствует аллелю A в положении 225340931;
(v) SM2973, расположенного на хромосоме 5 маиса, который соответствует аллелю G в положении 159121201;
(vi) SM2980, расположенного на хромосоме 9 маиса, который соответствует аллелю C в положении 12104936;
(vii) SM2982, расположенного на хромосоме 9 маиса, который соответствует аллелю A в положении 133887717; или
(viii) SM2984, расположенного на хромосоме 10 маиса, который соответствует аллелю G в положении 4987333; и
c) отбора первого растения маиса на основании присутствия молекулярного маркера, выявленного на b);
d) скрещивания растения маиса из c) со вторым растением маиса, не содержащим в своем геноме молекулярного маркера, выявленного в первом растении маиса; и
e) получения растения-потомка от скрещивания на d), что приводит к растению маиса, характеризующемуся повышенной урожайностью в условиях засухи и/или повышенной урожайностью, по сравнению с контрольным растением.
2. Способ по п. 1, где указанный молекулярный маркер локализован в пределах хромосомного интервала, фланкированного и включающего любое из:
a. IIM56014 и IIM48939 на хромосоме 1 маиса, расположенных в физических положениях пар оснований 248150852-296905665,
b. IIM39140 и IIM40144 на хромосоме 3 маиса, расположенных в физических положениях пар оснований 201538048-230992107,
c. IIM6931 и IIM7657 на хромосоме 9 маиса, расположенных в физических положениях пар оснований 121587239-145891243,
d. IIM40272 и IIM41535 на хромосоме 2 маиса, расположенных в физических положениях пар оснований 1317414-36929703,
e. IIM25303 и IIM48513 на хромосоме 5 маиса, расположенных в физических положениях пар оснований 139231600-183321037,
f. IIM4047 и IIM4978 на хромосоме 9 маиса, расположенных в физических положениях пар оснований 405220-34086738, или
g. IIM19 и IIM818 на хромосоме 10 маиса, расположенных в физических положениях пар оснований 1285447-29536061.
3. Способ по пп. 1, 2, где указанный молекулярный маркер локализован в пределах хромосомного интервала, включающего любое из:
a. хромосомного интервала на хромосоме 1 маиса, определяемого от положения пары оснований 272937470 до положения пары оснований 272938270 включительно;
b. хромосомного интервала на хромосоме 2 маиса, определяемого от положения пары оснований 12023306 до положения пары оснований 12024104 включительно;
c. хромосомного интервала на хромосоме 3 маиса, определяемого от положения пары оснований 225037202 до положения пары оснований 225038002 включительно;
d. хромосомного интервала на хромосоме 3 маиса, определяемого от положения пары оснований 225340531 до положения пары оснований 225341331 включительно;
e. хромосомного интервала на хромосоме 5 маиса, определяемого от положения пары оснований 159, 120, 801 до положения пары оснований 159, 121, 601 включительно;
f. хромосомного интервала на хромосоме 9 маиса, определяемого от положения пары оснований 12104536 до положения пары оснований 12105336 включительно;
g. хромосомного интервала на хромосоме 9 маиса, определяемого от положения пары оснований 225343590 до положения пары оснований 225340433 включительно; или
h. хромосомного интервала на хромосоме 10 маиса, определяемого от положения пары оснований 14764415 до положения пары оснований 14765098 включительно.
4. Способ по пп. 1-3, где выявленный молекулярный маркер является тесно ассоциированным с присутствием любого из следующих генов, кодирующих белок, содержащий любую из SEQ ID No: 9-16.
5. Способ по п. 4, где ген содержит любую из нуклеотидных последовательностей под SEQ ID No: 1-8.
6. Способ по пп. 1-5, где выявленный молекулярный маркер представляет собой любой аллель или тесно ассоциированный аллель, перечисленный в таблицах 1-7.
7. Способ по пп. 1-6, где выявление предусматривает: a) смешивание праймера для амплификации или пары праймеров для амплификации с нуклеиновой кислотой, выделенной из растения маиса или идиоплазмы маиса, где праймер или пара праймеров являются комплементарными или частично комплементарными по меньшей мере части маркерного локуса и способны инициировать полимеризацию ДНК с помощью ДНК-полимеразы при применении нуклеиновой кислоты маиса в качестве матрицы; и b) удлинение праймера или пары праймеров в реакции полимеризации ДНК, в которую включены ДНК-полимераза и нуклеиновая кислота, являющаяся матрицей, с образованием по меньшей мере одного информативного фрагмента, где информативный фрагмент содержит любой из маркеров, перечисленных в таблицах 1-7.
8. Способ по п. 7, где информативный фрагмент содержит любую из следующих SEQ ID No: 17-24.
| СПОСОБЫ ПОЛУЧЕНИЯ РАСТЕНИЙ С УЛУЧШЕННЫМ РОСТОМ В УСЛОВИЯХ ОГРАНИЧЕНИЯ УРОВНЯ АЗОТА | 2004 |
|
RU2333245C2 |
| БИОЛОГИЧЕСКИЙ ДНК МАРКЕР ДЛЯ ОПРЕДЕЛЕНИЯ СОРТОВ КАРТОФЕЛЯ, НАБОР И СПОСОБ СОРТОВОЙ ИДЕНТИФИКАЦИИ КАРТОФЕЛЯ | 2009 |
|
RU2413774C1 |
| WO 2005103301 A2, 03.11.2005. | |||
Авторы
Даты
2021-11-01—Публикация
2016-12-14—Подача