Изобретение относится к биотехнологии, генетической и белковой инженерии, и касается нового рекомбинантного штамма бактерий Escherichia coli (E. coli), который может быть использован для получения химерного рекомбинантного белка, состоящего из формамидопиримидин-ДНК-гликозилазы E. coli (Fpg) и усиленного зеленого флуоресцентного белка (EGFP) и способного к ковалентной сшивке с ДНК под действием боргидридов или цианоборгидридов щелочных металлов.
ДНК во всех живых клетках постоянно подвержена опасности повреждения. Эндогенные и экзогенные факторы - реакционноспособные побочные продукты окислительного метаболизма, спонтанный гидролиз, ксенобиотики, ультрафиолетовое и ионизирующее излучение и т. д. - порождают до 100 тыс. млн повреждений ДНК на клетку человека в день. Повреждения могут вести к мутациям и гибели клеток. На уровне человеческого организма это вносит большой вклад в развитие рака, нейродегенеративных и аутоиммунных заболеваний и в старение (1). Как правило, целостность ДНК поддерживается системой ферментативных путей, известных под общим названием репарации ДНК. Тем не менее, в геноме существует фоновый уровень повреждений ДНК, а его повышение является маркером генотоксического стресса. Типичным маркером повреждений ДНК служит, например, 8-оксогуанин (oxoG) - наиболее часто возникающее окисленное пуриновое основание, уровень которого резко повышается при окислительном стрессе, связанном с разнообразными патологическими процессами (2).
Среди различных типов повреждений ДНК наиболее распространены малые повреждения, незначительно искажающие структуру. Этот класс включает в себя дезаминированные, окисленные и алкилированные азотистые основания и апурин-апиримидиновые (АП-) сайты. В большинстве своем эти повреждения либо порождают мутации, заставляя клетку включать неправильные нуклеотиды в ДНК, либо цитотоксичны в результате блокирования репликации или транскрипции. Характерные для некоторых поврежденных оснований мутационные спектры, например, мутации G→T, вызванные окисленным азотистым основанием 8-оксогуанином (oxoG), обнаруживаются во многих опухолях человека (3). Исправление таких повреждений начинается с действия ферментов, принадлежащих к классу ДНК-гликозилаз, которые узнают поврежденные основания и удаляют их из ДНК.
Бактериальный фермент формамидопиримидин-ДНК-гликозилаза (Fpg) специфично узнает oxoG и некоторые другие окисленные пуриновые основания. В ходе реакции он образует с ДНК ковалентный интермедиат, который может быть восстановлен под действием донора гидрид-ионов, например, боргидридов или цианоборгидридов щелочных металлов. Восстановление протекает количественно и, как можно видеть из установленных третичных структур, не денатурирует белок (4).
Учитывая роль повреждений ДНК в молекулярной патологии, методы их обнаружения имеют первостепенное значение. Способность оценивать генотоксическую нагрузку становятся все более полезными в области эпидемиологии, оценки риска для здоровья, и клинического прогноза. Развитие надежных и удобных способов мониторинга образования поврежденных оснований in situ или в режиме иммуноферментного анализа было бы крайне полезно как для клинической медицины, так и для фундаментальной науки. Однако методы на основе антител, специфичных к повреждениям ДНК, наименее чувствительны и наименее специфичны. Это не удивительно, так как повреждениям приходится конкурировать за связывание антител с огромным избытком неповрежденной ДНК. С другой стороны, высокая специфичность ДНК-гликозилаз основана не на специфичности связывания с субстратом, а на специфичности каталитического процесса. Таким образом ДНК-гликозилазы, способные к ковалентной сшивке с ДНК в ходе удаления поврежденного основания, могут быть использованы как селективные распознающие модули в системах количественного анализа повреждений ДНК. В качестве сигнального модуля в таких системах могут быть использованы флуоресцентные белки, например, EGFP, присутствующие в виде домена в химерном белке с ДНК-гликозилазой.
На сегодня аналогов штаммов-продуцентов химерного белка формамидопиримидин-ДНК-гликозилазы с флуоресцентным репортерным белком, а также их аналогов, включающих иные ДНК-гликозилазы не обнаружено.
Задачей изобретения является создание рекомбинантного штамма - продуцента химерного белка формамидопиримидин-ДНК-гликозилазы с флуоресцентным репортерным белком.
Технический результат изобретения заключается в получении рекомбинантного штамма-продуцента, обеспечивающего получение химерного рекомбинантного белка Fpg-(G4S)3-TEVs-EGFP, способного к образованию ДНК-белковой сшивки.
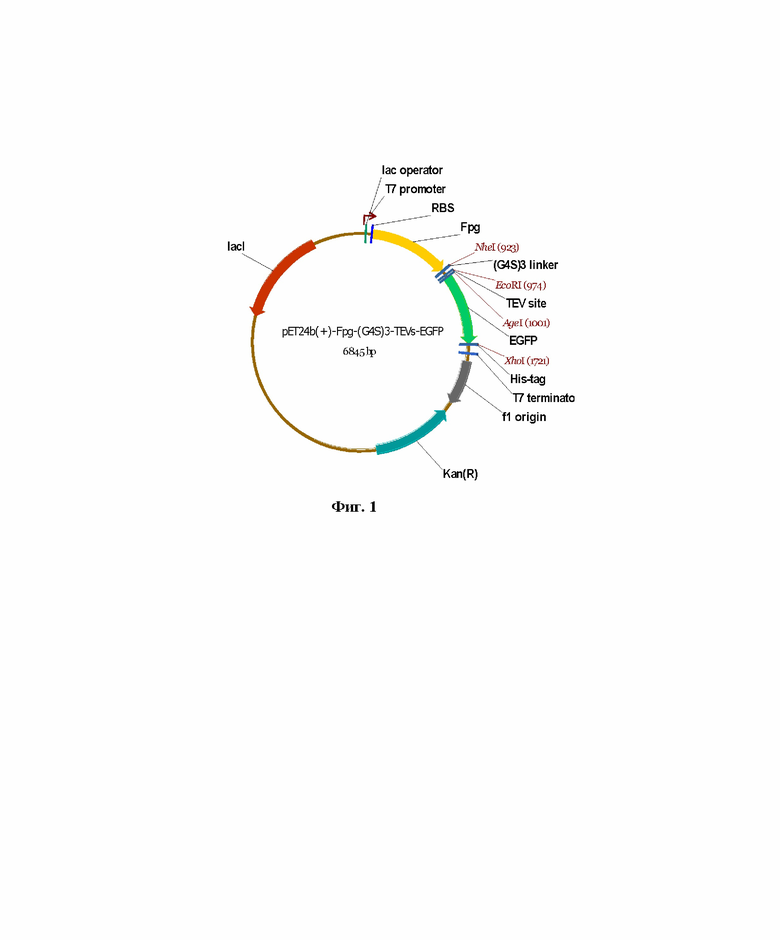
Поставленная задача достигается созданием рекомбинантного штамма E. coli BL21(DE3)/pET24b(+)-Fpg-(G4S)3-TEVs-EGFP - продуцента химерного рекомбинантного белка, состоящего из формамидопиримидин-ДНК-гликозилазы E. coli (Fpg) и усиленного зеленого флуоресцентного белка (EGFP), полученного трансформацией культуры клеток E. coli BL21(DE3) (генотип str. B F- ompT gal dcm lon hsdSB(rB-mB-) λ(DE3 [lacI lacUV5-T7p07 ind1 sam7 nin5]) [malB+]K-12(λS) pLysS(CamR)) рекомбинантной плазмидной ДНК pET24b(+)-Fpg-(G4S)3-TEVs-EGFP, сконструированной на основе вектора pET24b(+), несущего фрагмент гена, кодирующего химерный рекомбинантный белок, состоящий из формамидопиримидин-ДНК-гликозилазы E. coli (Fpg) и усиленного зеленого флуоресцентного белка (EGFP) - Fpg-(G4S)3-TEVs-EGFP. На фиг. 1 представлена физическая карта плазмиды pET24b-Fpg(G4S)3TEV-EGFP.
Культурально-морфологические особенности штамма E. coli BL21(DE3)/pET24b(+)-Fpg-(G4S)3-TEVs-EGFP: грамотрицательные прямые палочки, размером 1,1-1,5×2,0-3,0 мкм, одиночные, спор и капсул не образуют. Каталазоположительные. Оксидазоотрицательные. Факультативные анаэробы. Клетки хорошо растут на простых питательных средах, содержащих и не содержащих канамицин, например, на среде LB (питательная среда Lisogeny Broth). На агаризованной среде - колонии гладкие, круглые, слабо выпуклые, с ровным краем. В жидких средах образуют равномерную светорассеивающую суспензию, при хранении без перемешивания оседают на дно. Клетки растут в интервале температур от 8°C до 43°C, интервал для наиболее эффективного культивирования - 28-38°C, оптимум роста при 37°C. Оптимальный интервал pH для культивирования pH 5-7. Изначальный штамм E. coli BL21(DE3) не проявляет устойчивость к каким-либо антибиотикам.
Характеристики полезного вещества, синтезируемого штаммом: химерный рекомбинантный белок, состоящий из формамидопиримидин-ДНК-гликозилазы E. coli (Fpg) длиной 269 аминокислотных остатков и усиленного зеленого флуоресцентного белка (EGFP) длиной 233 аминокислотных остатков, между белками в молекуле химерного рекомбинантного белка находятся: небольшой линкер (2 аминокислоты), линкер (GlyGlyGlyGlySer)3 (15 аминокислот), небольшой линкер (2 аминокислоты), сайт посадки для TEV-протеазы (7 аминокислот), небольшой линкер (7 аминокислот), а на С-конце химерного рекомбинантного белка находится линкер (2 аминокислоты) и гистидиновая метка (6 аминокислот) .
Продуктивность созданного штамма - химерный рекомбинантный белок Fpg-(G4S)3-TEVs-EGFP, составляет не менее 20 % белка клеточного лизата при культивировании в жидкой питательной среде LB при 37°С, 250 об/мин, в условиях индукции 100 мкМ ИПТГ (изопропил-β-D-1-тиогалактопиранозид) в течение 3 часов с добавлением антибиотика и канамицина (100 мкг/мл).
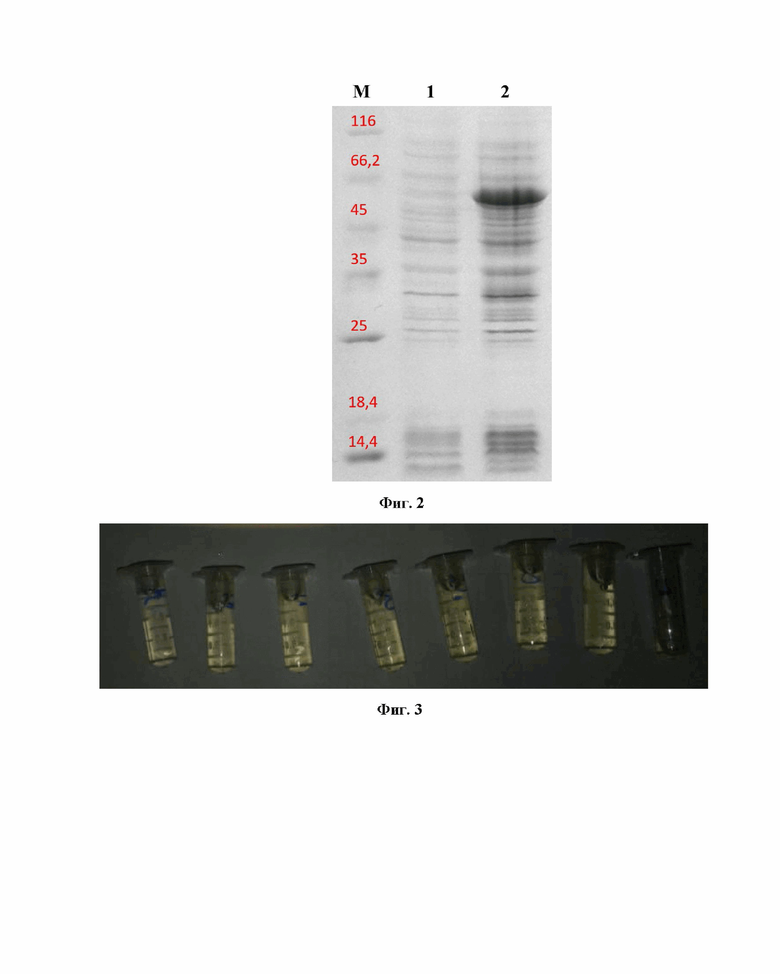
На фиг. 2 приведена электрофореграмма после разделения клеточных лизатов клеток E. coli BL21(DE3)/pET24b(+)-Fpg-(G4S)3-TEVs-EGFP при культивировании в условиях индукции синтеза белка при помощи ИПТГ и без индукции; где М - белковый маркер подвижности 14,4-116 кДа (ThermoScientific); 1 - контрольная неиндуцированная культура, 3 ч после индукции 100 мкМ ИПТГ.
Сущность изобретения поясняется следующими конкретными примерами получения штамма E. coli BL21(DE3)/pET24b(+)-Fpg-(G4S)3-TEVs-EGFP.
Пример 1. Создание генетической конструкции, обеспечивающей синтез химерного рекомбинантного белка Fpg-(G4S)3-TEVs-EGFP в клетках Е. coli.
Клонирование гена EGFP и сайта расщепления TEV-протеазой в ранее полученный экспрессионный вектор, содержащей ген Fpg, а также линкер (GlyGlyGlyGlySer)3, осуществлялось по сайтам рестрикции EcoR I и Xho I: Для этого проводилась амплификация гена EGFP из имеющейся в лаборатории плазмиды с праймерами (табл. 1), содержащими последовательности сайтов рестрикции EcoR I, AgeI и расщепления TEV-протеазой с 5’-конца, и сайта рестрикции Xho I с 3’-конца. Продукт амплификации и вектор подвергались обработке эндонуклеазами рестрикции EcoR I и Xho I, рестрикционные смеси очищались. Затем проводилось лигирование фрагментов при +4°С в течении 16 ч. После чего полученной лигазной смесью трансформировались клетки штамма Escherichia coli XL1 Blue. Положительные по данным метода ПЦР колонии были пересеяны в ночные культуры, из ночных культур выделялась плазмидная ДНК для подтверждения последовательности секвенированием по Сэнгеру. Последовательность нуклеотидов соотносится с необходимой аминокислотной последовательностью химерного белка Fpg-(G4S)3-TEVs-EGFP.
На фиг. 1 представлена физическая карта плазмиды pET24b(+)-Fpg-(G4S)3-TEVs-EGFP, содержащая ориджин репликации f1, промотор фага T7, RBS-сайт посадки рибосомы, сайты рестрикции Nhe I, EcoR I, Age I и Xho I, встроенный ген, кодирующий химерный белок Fpg-(G4S)3-TEVs-EGFP, состоящий из ДНК-гликозилазы Fpg из бактерии Escherichia coli и усиленного зеленого флуоресцентного белка EGFP, а также линкера (GlyGlyGlyGlySer)3 и сайт расщепления TEV-протеазой между Fpg и EGFP, гистидиновую метку His-tag, терминатор фага T7, ген устойчивости к канамицину Kan (R), лактозный репрессор lacI и лактозный оператор.
Полученная плазмида pET24b(+)-Fpg-(G4S)3-TEVs-EGFP обеспечивает синтез в клетках Escherichia coli химерного рекомбинантного белка Fpg-(G4S)3-TEVs-EGFP, содержащего ДНК-гликозилазы Fpg из бактерии Escherichia coli и усиленного зеленого флуоресцентного белка EGFP, а также линкера (GlyGlyGlyGlySer)3 и сайт расщепления TEV-протеазой между Fpg и EGFP, гистидиновую метку His-tag, предназначенную для последующей очистки рекомбинантного белка Fpg-(G4S)3-TEVs-EGFP c помощью металлохелатной хроматографии.
Нуклеотидная последовательность рекомбинантной плазмиды pET24b(+)-Fpg-(G4S)3-TEVs-EGFP (SEQ ID NO 1) представлена в перечне последовательностей.
Пример 2. Получение штамма-продуцента химерного рекомбинантного белка, состоящего из формамидопиримидин-ДНК-гликозилазы E. coli (Fpg) и усиленного зеленого флуоресцентного белка (EGFP) и исследование его продуктивности.
Полученной плазмидой pET24b-Fpg(G4S)3TEV-EGFP были трансформированы клетки E. coli штамма BL21(DE3), содержащие в своем геноме ген, кодирующий ДНК-зависимую РНК-полимеразу фага Т7, под контролем промотора lacUV5, индуцируемого лактозой или ИПТГ. Кроме того, клетки E. coli BL21(DE3) дефектны по генам протеаз Lon и ompT, что снижает деградацию гетерологичных белков.
В результате был получен штамм E. coli BL21(DE3)/pET24b-Fpg(G4S)3TEV-EGFP - продуцент химерного рекомбинантного белка Fpg-(G4S)3-TEVs-EGFP, состоящего из формамидопиримидин-ДНК-гликозилазы E. coli (Fpg) и усиленного зеленого флуоресцентного белка (EGFP).
Для поддержания полученного штамма-продуцента белка Fpg-(G4S)3-TEVs-EGFP использовали плотную агаризованную LB-среду, содержащую 100 мкг/мл канамицина.
Продуктивность полученного штамма-продуцента изучали путем культивирования клеток в среде LB, в термостатированном шейкере роторного типа при температуре 37°С, скорости вращения платформы 250 об/мин до оптической плотности А600 = 0,8. После этого индуцировали синтез целевого белка добавлением ИПТГ до конечной концентрации 100 мкМ. В качестве контроля использовали культуру без добавления ИПТГ. Образцы собранной центрифугированием биомассы клеток лизировали при помощи ультразвукового разрушения и анализировали методом электрофореза в 12 %-полиакриламидном геле в системе Лэммли в денатурирующих условиях. Результат представлен на фиг. 2, где М - белковый маркер подвижности 14,4-116 кДа (ThermoScientific); 1 - контрольная неиндуцированная культура, 3 ч после индукции 100 мкМ ИПТГ. В результате выявлено, что индукция 100 мкМ ИПТГ культуры клеток E. coli приводит к синтезу белка с молекулярной массой 60 кДа, что соответствует ожидаемой молекулярной массе для химерного рекомбинантного белка Fpg-(G4S)3-TEVs-EGFP с гистидиновой меткой. Количество рекомбинантного белка составило не менее 20 % от клеточного лизата при культивировании в условиях индукции.
Аминокислотная последовательность химерного рекомбинантного белка Fpg-(G4S)3-TEVs-EGFP (SEQ ID NО: 2) представлена в перечне последовательностей.
Пример 3. Очистка химерного рекомбинантного белка Fpg-(G4S)3-TEVs-EGFP и изучение его активности.
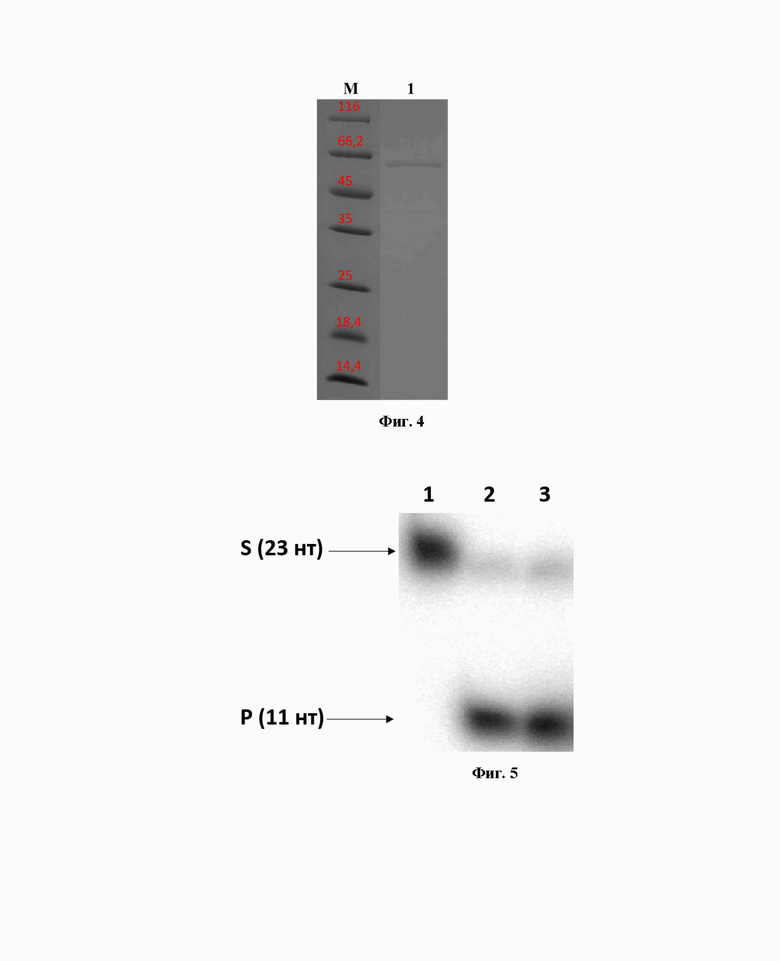
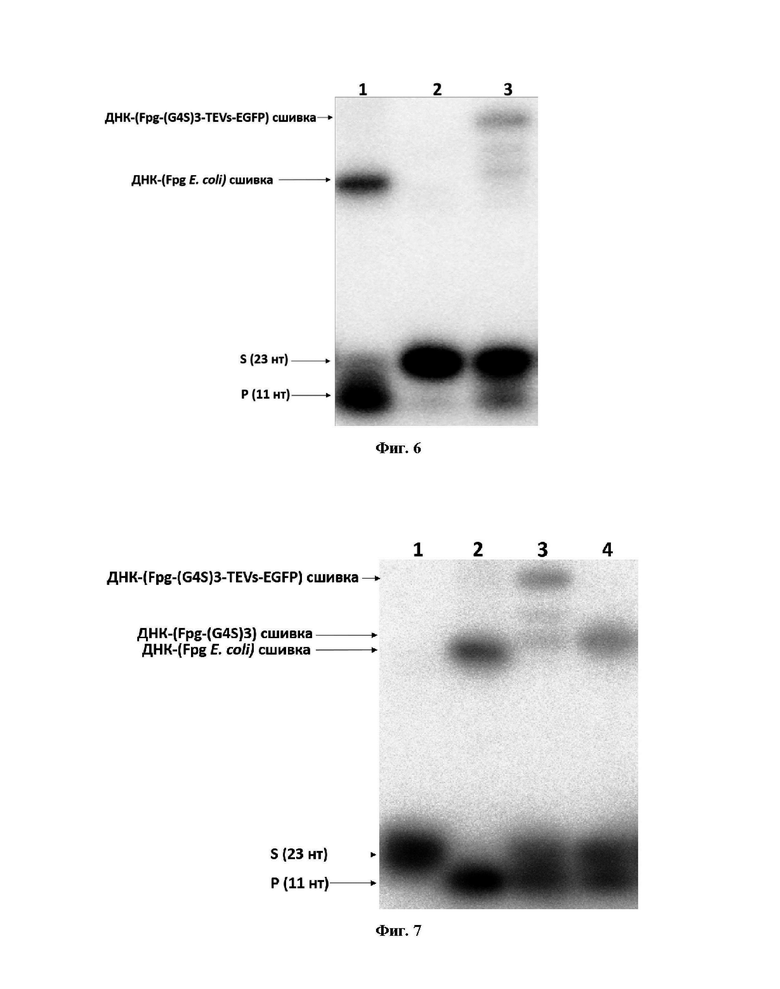
Очистку химерного рекомбинантного белка проводили в две стадии. Первая стадия включала в себя аффинную хроматографию на металлхелатном агарозном сорбенте, модифицированном иминодиуксусной кислотой, заряженной ионами Ni2+. Вторая стадия очистки представляла собой ионообменную хроматографию на сорбенте Heparin HP-сефарозе. На фиг. 3 представлено фото фракций после хроматографии. После этого белок диализовали против буфера, содержащего 50 %-ный глицерин, и хранили при -20°C. Результат двухстадийной очистки Fpg-(G4S)3-TEVs-EGFP представлен на фиг. 4, где М - маркер подвижности 14,4-116 кДа (ThermoScientific); 1 - очищенный рекомбинантный белок Fpg-(G4S)3-TEVs-EGFP. Как видно из фиг. 4 препарат белка Fpg-(G4S)3-TEVs-EGFP является практически гомогенным, и его чистота составляет не менее 90 %.
Для исследования активности очищенного белка Fpg-(G4S)3-TEVs-EGFP in vitro в качестве субстрата использовали двуцепочечный олигодезоксирибонуклеотид длиной 23 пары оснований, содержащий окислительное повреждение oxoG напротив C и радиоактивную метку на 5’-конце в конечной концентрации 50 нМ. Реакцию проводили при 37 С в течение 10 мин. Продукты реакции анализировали электрофорезом в 20 %-полиакриламидном геле в денатурирующих условиях с последующим радиолюминесцентным сканированием. Результат эксперимента представлен на фиг. 5, где 1 - субстрат без фермента; 2 - положительный контроль с Fpg E. coli wt, с конечной концентрацией фермента 250 нм; 3 - реакция с Fpg-(G4S)3-TEVs-EGFP с конечной концнтрацией фермета 250 нм; S-субстрат, P - продукт реакции. Из фиг. 5 видно, что очищенный в рекомбинантном виде белок Fpg-(G4S)3-TEVs-EGFP проявлял активность в отношении субстрата, содержащего типичное окислительное повреждение oxoG напротив C на уровне Fpg E. coli wt, что говорит о том, что гликозилазная активность сохраняется в химерном рекомбинантом белке Fpg-(G4S)3-TEVs-EGFP.
Также проанализировали возможность образования ДНК-белковой сшивки. В качестве субстрата использовали двуцепочечный олигодезоксирибонуклеотид длиной 23 пары оснований, содержащий апурин-апиримидиновый сайт (АП-сайт) напротив C и радиоактивную метку на 5’-конце в конечной концентрации 50 нМ. Для образования ДНК-белковой сшивки в реакционную смесь добавляли NaBH4 до конечной концентрации 500 мМ. Реакцию проводили при 37 С в течение 10 мин. Продукты реакции анализировали электрофорезом в системе Лэммли в 12 %-полиакриламидном геле с последующим радиолюминесцентным сканированием. Результат эксперимента представлен на фиг. 6, где 1 - реакция с Fpg E. coli wt, с конечной концентрацией фермата 250 нм; 2 - субстрат без фермента; 3 - реакция с Fpg-(G4S)3-TEVs-EGFP, с конечной концентрацией фермата 250 нм; S-субстрат, P - продукт реакции. Из фиг. 6 видно, что очищенный в рекомбинантном виде белок Fpg-(G4S)3-TEVs-EGFP образует сшивку хуже, чем Fpg E. coli wt, что может быть связано с увеличившейся массой молекулы фермента, из-за чего оборотность фермента могла уменьшиться. Видно, что достаточно много субстрата не образовало ковалентную сшивку с химерным рекомбинантным белком из-за того, что альдегидн АП-сайт восстановился быстрей, чем молекула химерного белка успела настигнуть повреждение.
Для исследования доступности сайта расщепления TEV протеазой проанализировали расщепление ДНК-белковой сшивки с химерным рекомбинантным белком Fpg-(G4S)3-TEVs-EGFP TEV-протеазой. В качестве субстрата получали ДНК-белковую сшивку с Fpg E. coli и Fpg-(G4S)3-TEVs-EGFP как описано выше. Далее, к полученной ДНК-белковой сшивке с химерным белком добавляли TEV-протеазу до конечной концентрации 300 нМ. Реакцию проводили при 25С 1 ч. Продукты реакции анализировали электрофорезом в системе Лэммли в 12 %-полиакриламидном геле с последующим радиолюминесцентным сканированием. Результат эксперимента представлен на фиг. 7, где 1 - субстрат без фермента; 2 - реакция с Fpg E. coli wt, с конечной концентрацией фермента 250 нм; 3 - реакция с Fpg-(G4S)3-TEVs-EGFP, с конечной концентрацией фермента 250 нм; 4 - реакция ДНК-(Fpg-(G4S)3-TEVs-EGFP) сшивки с TEV-протеазой, с конечной концентрацией фермента 300 нм; S-субстрат, P - продукт реакции. Из фиг. 7 видно, что полученный после протеолиза продукт - ДНК-(Fpg-(G4S)3) сшивка находится чуть выше, чем ДНК-(Fpg E. coli) из-за наличия линкера (GlyGlyGlyGlySer)3. Важно, что сайт расщепления TEV-протеазой находится в доступности к действию TEV-протеазы, притом, что это не влияет на активность домена Fpg в химерном рекомбинантном белке Fpg-(G4S)3-TEVs-EGFP.
Пример 4. Проверка флуоресценции химерного рекомбинантного белка Fpg-(G4S)3-TEVs-EGFP.
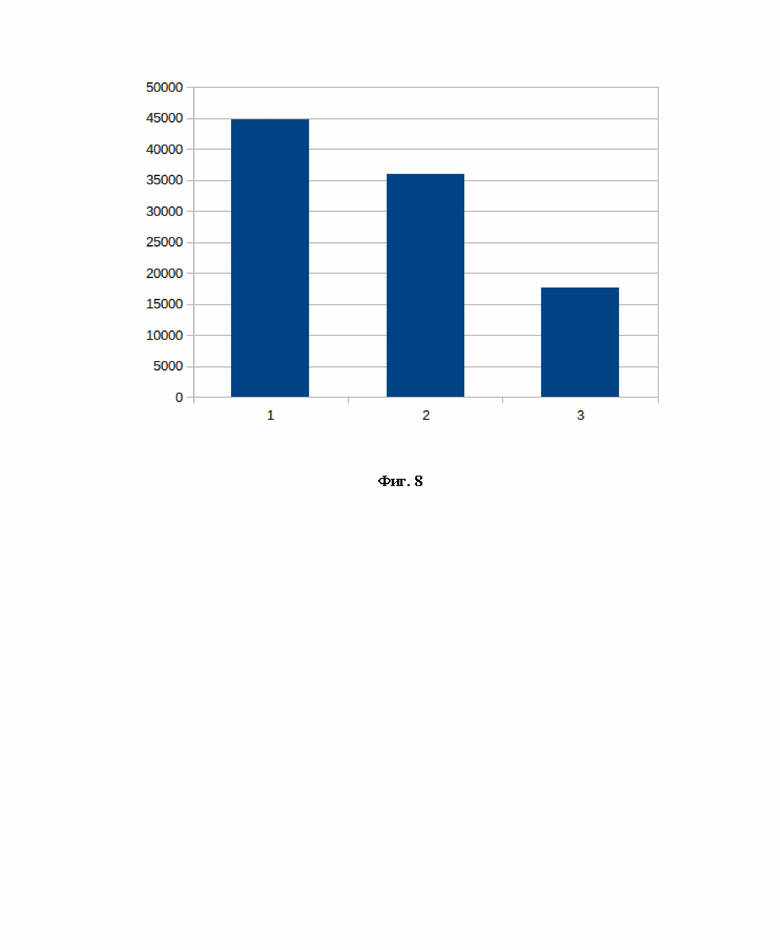
Для исследования флуоресценции сравнивались флуоресцентные спектры химерного белка Fpg-(G4S)3-TEVs-EGFP, реакционной смеси для образования ДНК-(Fpg-(G4S)3-TEVs-EGFP) сшивки без добавления NaBH4 и реакционной смеси для образования ДНК-( Fpg-(G4S)3-TEVs-EGFP) сшивки с добавлением NaBH4. ДНК-белковую сшивку с Fpg E. coli и Fpg-(G4S)3-TEVs-EGFP получали как описано выше. Далее, отбирались по 50 мкл реакционные смеси и анализировались на флуориметре. На фиг. 8 показаны результаты флуорометрии, где 1 - флуоресценция химерного белка Fpg-(G4S)3-TEVs-EGFP, 2 - флуоресценция реакционной смеси для образования ДНК-(Fpg-(G4S)3-TEVs-EGFP) сшивки без добавления NaBH4, флуоресценция реакционной смеси для образования ДНК-(Fpg-(G4S)3-TEVs-EGFP) сшивки с добавлением NaBH4. Из фиг. 8 видно, что флуоресценция снижается примерно на 20%, а при обработке реакционной смеси NaBH4 флуоресценция снижается примерно в 2 раза относительно флуоресценции реакционной смеси для образования ДНК-( Fpg-(G4S)3-TEVs-EGFP) без добавления NaBH4, но осталась на детектируемом уровне, что говорит о том, что химерный рекомбинантный белок Fpg-(G4S)3-TEVs-EGFP можно использовать как для качественного, так и для количественного анализа количества окислительных или апуринизированных повреждений ДНК.
Таким образом, создан штамм бактерий Escherichia сoli BL21(DE3)/pET24b-Fpg(G4S)3TEV-EGFP, являющийся продуцентом химерного рекомбинантного белка Fpg-(G4S)3-TEVs-EGFP, содержащего ДНК-гликозилазу Fpg из бактерии Escherichia coli и усиленного зеленого флуоресцентного белка EGFP, а также линкера (GlyGlyGlyGlySer)3, сайта расщепления TEV-протеазой между Fpg и EGFP и гистидиновую метку His-tag. Рекомбинантный белок Fpg-(G4S)3-TEVs-EGFP может служить эффективным инструментом для количественного анализа окислительных и апуринизированных повреждений ДНК.
Источники информации
1. Schumacher B., Pothof J., Vijg J., Hoeijmakers J.H.J. The central role of DNA damage in the ageing process // Nature. - 2021. - V. 592. - No. 7856. - P. 695-703.
2. Jacob K.D., Hooten N.N., Trzeciak A.äR., Evans M.K. Markers of oxidant stress that are clinically relevant in aging and age-related disease // Mech. Ageing Dev. - 2013. - V. 134. - No. 3-4. - P. 139-157.
3. Alexandrov L.B., Nik-Zainal S., Wedge D.C., Aparicio S.A.J.R., Behjati S., Biankin A.V., Bignell G.R., Bolli N., Borg A., Børresen-Dale A.-L., Boyault S., Burkhardt B., Butler A.P., Caldas C., Davies H.R., Desmedt C., Eils R., Eyfjörd J.E., Foekens J.A., Greaves M., Hosoda F., Hutter B., Ilicic T., Imbeaud S., Imielinski M., Jäger N., Jones D.T.W., Jones D., Knappskog S., Kool M., Lakhani S.R., López-Otín C., Martin S., Munshi N.C., Nakamura H., Northcott P.A., Pajic M., Papaemmanuil E., Paradiso A., Pearson J.V., Puente X.S., Raine K., Ramakrishna M., Richardson A.L., Richter J., Rosenstiel P., Schlesner M., Schumacher T.N., Span P.N., Teague J.W., Totoki Y., Tutt A.N.J., Valdés-Mas R., van Buuren M.M., van ’t Veer L., Vincent-Salomon A., Waddell N., Yates L.R., Australian Pancreatic Cancer Genome Initiative, ICGC Breast Cancer Consortium, ICGC MMML-Seq Consortium, ICGC PedBrain, Zucman-Rossi J., Futreal P.A., McDermott U., Lichter P., Meyerson M., Grimmond S.M., Siebert R., Campo E., Shibata T., Pfister S.M., Campbell P.J., Stratton M.R. Signatures of mutational processes in human cancer // Nature. - 2013. - V. 500. - No. 7463. - P. 415-421.
4. Zharkov D.O., Shoham G., Grollman A.P. Structural characterization of the Fpg family of DNA glycosylases // DNA Repair. - 2003. - V. 2. - No. 8. - P. 839-862.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3"
fileName="FPG-EGFP_protein_final.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-11-27">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate></FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Новосибирский национальный
исследовательский государственный университет, Институт химической
биологии и фундаментальной медицины Сибирского отделения Российской
академии наук</ApplicantName>
<ApplicantNameLatin>Novosibirsk State University, Institute of
Chemical Biology and Fundamental Medicine, Siberian Branch of the
Russian Academy of Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru">Рекомбинантный штамм бактерий
Escherichia coli - продуцент химерного белка
формамидопиримидин-ДНК-гликозилазы с флуоресцентным репортерным
белком</InventionTitle>
<SequenceTotalQuantity>2</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>6845</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..6845</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agatctcgatcccgcgaaattaatacgactcactataggggaattgtga
gcggataacaattcccctctagaaataattttgtttaacatttaaatccagaaggagatataactatgcc
tgaattacccgaagtagaaaccagccgccgcggcatagaaccgcatctcgttggtgcaaccattcttcat
gcagtggtgcgcaacggacgcttgcgctggccggtttcagaagagatctaccgtttaagcgaccaaccag
tgcttagcgtgcagcggcgggctaaatatctgctgctggagctgcctgagggctggattatcattcattt
agggatgtctggcagcctgcgcatccttccagaagaacttccccctgaaaagcatgaccatgtggatttg
gtgatgagcaacggcaaagtgctgcgctacaccgatccgcgccgctttggtgcctggctgtggaccaaag
agctggaagggcataatgtgctgacccatcttggaccggagccgcttagcgacgatttcaatggtgagta
tctgcatcagaagtgcgcgaagaaaaaaacggcgattaaaccgtggctgatggataacaagctggtggta
ggggtagggaatatctatgccagcgaatcactgtttgcggcggggatccatccggatcggctggcgtcat
cactgtcgctggcagagtgtgaattgttagctcgggtgattaaagcggtgttgctgcgttcgattgagca
gggtggtacaacgctgaaagattttctgcaaagtgatggtaaaccgggctatttcgctcaggaattgcag
gtttacgggcgaaaaggtgagccgtgtcgggtgtgcggtacgccgattgtggcgactaaacatgcgcagc
gggcaacgttttattgtcggcagtgccagaaggctagcggaggaggaggaagtggaggaggaggaagtgg
aggaggaggaagtgaattcgagaatctgtacttccagtctaccggtgtgagcaagggcgaggagctgttc
accggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaagttcagcgtgtccggcg
agggcgagggcgatgccacctacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgt
gccctggcccaccctcgtgaccaccctgacctacggcgtgcagtgcttcagccgctaccccgaccacatg
aagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaagg
acgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgagct
gaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctggagtacaactacaacagccac
aacgtctatatcatggccgacaagcagaagaacggcatcaaggtgaacttcaagatccgccacaacatcg
aggacggcagcgtgcagctcgccgaccactaccagcagaacacccccatcggcgacggccccgtgctgct
gcccgacaaccactacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatg
gtcctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagctcgagcacc
accaccaccaccactgagatccggctgctaacaaagcccgaaaggaagctgagttggctgctgccaccgc
tgagcaataactagcataaccccttggggcctctaaacgggtcttgaggggttttttgctgaaaggagga
actatatccggattggcgaatgggacgcgccctgtagcggcgcattaagcgcggcgggtgtggtggttac
gcgcagcgtgaccgctacacttgccagcgccctagcgcccgctcctttcgctttcttcccttcctttctc
gccacgttcgccggctttccccgtcaagctctaaatcgggggctccctttagggttccgatttagtgctt
tacggcacctcgaccccaaaaaacttgattagggtgatggttcacgtagtgggccatcgccctgatagac
ggtttttcgccctttgacgttggagtccacgttctttaatagtggactcttgttccaaactggaacaaca
ctcaaccctatctcggtctattcttttgatttataagggattttgccgatttcggcctattggttaaaaa
atgagctgatttaacaaaaatttaacgcgaattttaacaaaatattaacgtttacaatttcaggtggcac
ttttcggggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccgctc
atgaattaattcttagaaaaactcatcgagcatcaaatgaaactgcaatttattcatatcaggattatca
ataccatatttttgaaaaagccgtttctgtaatgaaggagaaaactcaccgaggcagttccataggatgg
caagatcctggtatcggtctgcgattccgactcgtccaacatcaatacaacctattaatttcccctcgtc
aaaaataaggttatcaagtgagaaatcaccatgagtgacgactgaatccggtgagaatggcaaaagttta
tgcatttctttccagacttgttcaacaggccagccattacgctcgtcatcaaaatcactcgcatcaacca
aaccgttattcattcgtgattgcgcctgagcgagacgaaatacgcgatcgctgttaaaaggacaattaca
aacaggaatcgaatgcaaccggcgcaggaacactgccagcgcatcaacaatattttcacctgaatcagga
tattcttctaatacctggaatgctgttttcccggggatcgcagtggtgagtaaccatgcatcatcaggag
tacggataaaatgcttgatggtcggaagaggcataaattccgtcagccagtttagtctgaccatctcatc
tgtaacatcattggcaacgctacctttgccatgtttcagaaacaactctggcgcatcgggcttcccatac
aatcgatagattgtcgcacctgattgcccgacattatcgcgagcccatttatacccatataaatcagcat
ccatgttggaatttaatcgcggcctagagcaagacgtttcccgttgaatatggctcataacaccccttgt
attactgtttatgtaagcagacagttttattgttcatgaccaaaatcccttaacgtgagttttcgttcca
ctgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgc
tgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctaccaactcttt
ttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttagg
ccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgct
gccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcggt
cgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacct
acagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggc
agggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcctgtcg
ggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaa
cgccagcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttctttcctgcg
ttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgataccgctcgccgcagccgaa
cgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgcctgatgcggtattttctccttacgca
tctgtgcggtatttcacaccgcatatatggtgcactctcagtacaatctgctctgatgccgcatagttaa
gccagtatacactccgctatcgctacgtgactgggtcatggctgcgccccgacacccgccaacacccgct
gacgcgccctgacgggcttgtctgctcccggcatccgcttacagacaagctgtgaccgtctccgggagct
gcatgtgtcagaggttttcaccgtcatcaccgaaacgcgcgaggcagctgcggtaaagctcatcagcgtg
gtcgtgaagcgattcacagatgtctgcctgttcatccgcgtccagctcgttgagtttctccagaagcgtt
aatgtctggcttctgataaagcgggccatgttaagggcggttttttcctgtttggtcactgatgcctccg
tgtaagggggatttctgttcatgggggtaatgataccgatgaaacgagagaggatgctcacgatacgggt
tactgatgatgaacatgcccggttactggaacgttgtgagggtaaacaactggcggtatggatgcggcgg
gaccagagaaaaatcactcagggtcaatgccagcgcttcgttaatacagatgtaggtgttccacagggta
gccagcagcatcctgcgatgcagatccggaacataatggtgcagggcgctgacttccgcgtttccagact
ttacgaaacacggaaaccgaagaccattcatgttgttgctcaggtcgcagacgttttgcagcagcagtcg
cttcacgttcgctcgcgtatcggtgattcattctgctaaccagtaaggcaaccccgccagcctagccggg
tcctcaacgacaggagcacgatcatgcgcacccgtggggccgccatgccggcgataatggcctgcttctc
gccgaaacgtttggtggcgggaccagtgacgaaggcttgagcgagggcgtgcaagattccgaataccgca
agcgacaggccgatcatcgtcgcgctccagcgaaagcggtcctcgccgaaaatgacccagagcgctgccg
gcacctgtcctacgagttgcatgataaagaagacagtcataagtgcggcgacgatagtcatgccccgcgc
ccaccggaaggagctgactgggttgaaggctctcaagggcatcggtcgagatcccggtgcctaatgagtg
agctaacttacattaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagctgc
attaatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgccagggtggtttttcttttca
ccagtgagacgggcaacagctgattgcccttcaccgcctggccctgagagagttgcagcaagcggtccac
gctggtttgccccagcaggcgaaaatcctgtttgatggtggttaacggcgggatataacatgagctgtct
tcggtatcgtcgtatcccactaccgagatatccgcaccaacgcgcagcccggactcggtaatggcgcgca
ttgcgcccagcgccatctgatcgttggcaaccagcatcgcagtgggaacgatgccctcattcagcatttg
catggtttgttgaaaaccggacatggcactccagtcgccttcccgttccgctatcggctgaatttgattg
cgagtgagatatttatgccagccagccagacgcagacgcgccgagacagaacttaatgggcccgctaaca
gcgcgatttgctggtgacccaatgcgaccagatgctccacgcccagtcgcgtaccgtcttcatgggagaa
aataatactgttgatgggtgtctggtcagagacatcaagaaataacgccggaacattagtgcaggcagct
tccacagcaatggcatcctggtcatccagcggatagttaatgatcagcccactgacgcgttgcgcgagaa
gattgtgcaccgccgctttacaggcttcgacgccgcttcgttctaccatcgacaccaccacgctggcacc
cagttgatcggcgcgagatttaatcgccgcgacaatttgcgacggcgcgtgcagggccagactggaggtg
gcaacgccaatcagcaacgactgtttgcccgccagttgttgtgccacgcggttgggaatgtaattcagct
ccgccatcgccgcttccactttttcccgcgttttcgcagaaacgtggctggcctggttcaccacgcggga
aacggtctgataagagacaccggcatactctgcgacatcgtataacgttactggtttcacattcaccacc
ctgaattgactctcttccgggcgctatcatgccataccgcgaaaggttttgcgccattcgatggtgtccg
ggatctcgacgctctcccttatgcgactcctgcattaggaagcagcccagtagtaggttgaggccgttga
gcaccgccgccgcaaggaatggtgcatgcaaggagatggcgcccaacagtcccccggccacggggcctgc
caccatacccacgccgaaacaagcgctcatgagcccgaagtggcgagcccgatcttccccatcggtgatg
tcggcgatataggcgccagcaaccgcacctgtggcgccggtgatgccggccacgatgcgtccggcgtaga
ggatcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>543</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..543</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MPELPEVETSRRGIEPHLVGATILHAVVRNGRLRWPVSEEIYRLSDQPV
LSVQRRAKYLLLELPEGWIIIHLGMSGSLRILPEELPPEKHDHVDLVMSNGKVLRYTDPRRFGAWLWTKE
LEGHNVLTHLGPEPLSDDFNGEYLHQKCAKKKTAIKPWLMDNKLVVGVGNIYASESLFAAGIHPDRLASS
LSLAECELLARVIKAVLLRSIEQGGTTLKDFLQSDGKPGYFAQELQVYGRKGEPCRVCGTPIVATKHAQR
ATFYCRQCQKASGGGGSGGGGSGGGGSEFENLYFQSTGVSKGEELFTGVVPILVELDGDVNGHKFSVSGE
GEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKD
DGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIE
DGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYKLEHH
HHHH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к биотехнологии. Предложен рекомбинантный штамм бактерий Escherichia coli, являющийся продуцентом химерного белка формамидопиримидин-ДНК-гликозилазы (Fpg) с флуоресцентным репортерным белком (EGFP). Указанный штамм получен путем трансформации клеток Escherichia coli штамма BL21(DE3) плазмидой pET24b(+)-Fpg-(G4S)3-TEVs-EGFP, имеющей нуклеотидную последовательность SEQ ID NО: 1, сконструированной на основе вектора pET24b(+) и содержащей ориджин репликации f1, промотор фага T7, RBS-сайт посадки рибосомы, встроенный ген, кодирующий химерный рекомбинантный белок Fpg и EGFP, имеющий аминокислотную последовательность SEQ ID NО: 2, терминатор фага T7, ген устойчивости к канамицину Kan (R), лактозный репрессор lacI и лактозный оператор. Изобретение обеспечивает получение рекомбинантного штамма-продуцента, обеспечивающего получение химерного рекомбинантного белка формамидопиримидин-ДНК-гликозилазы (Fpg) с флуоресцентным репортерным белком (EGFP), способного к образованию ДНК-белковой сшивки. 8 ил., 4 пр.
Рекомбинантный штамм бактерий Escherichia coli – продуцент химерного белка формамидопиримидин-ДНК-гликозилазы (Fpg) с флуоресцентным репортерным белком (EGFP), полученный путем трансформации клеток Escherichia coli штамма BL21(DE3) плазмидой pET24b(+)-Fpg-(G4S)3-TEVs-EGFP, имеющей нуклеотидную последовательность SEQ ID NО: 1, сконструированной на основе вектора pET24b(+) и содержащей ориджин репликации f1, промотор фага T7, RBS-сайт посадки рибосомы, встроенный ген, кодирующий химерный рекомбинантный белок Fpg и EGFP, имеющий аминокислотную последовательность SEQ ID NО: 2, терминатор фага T7, ген устойчивости к канамицину Kan (R), лактозный репрессор lacI и лактозный оператор.
| ROPOLO M | |||
| ET AL | |||
| Accelerated repair and reduced mutagenicity of oxidative DNA damage in human bladder cells expressing the E | |||
| coli FPG protein | |||
| Int | |||
| J | |||
| Прибор для массовой выработки лекал | 1921 |
|
SU118A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Видоизменение пишущей машины для тюркско-арабского шрифта | 1923 |
|
SU25A1 |
| MINETTI C.A | |||
| ET AL | |||
| A continuous hyperchromicity assay to | |||
Авторы
Даты
2025-02-03—Публикация
2023-12-12—Подача