Область техники
Настоящее изобретение относится к области биотехнологии, в частности к системам управления экспрессией генов, системам создания синтетических генетических схем в живых клетках, системам адресной доставки молекул к областям генома, системам редактирования генома и эпигенома на основе нуклеотидной последовательности, кодирующей белки nCas9/dCas9, и нуклеотидных последовательностей, кодирующих молекулы РНК с самодимеризующимися РНК доменами, в качестве инструмента для повышения специфичности связывания CRISPR/nCas9 и CRISPR/dCas9 с целевыми последовательностями ДНК. Способ повышения специфичности связывания CRISPR/nCas9 и CRISPR/dCas9 комплексов, согласно настоящему изобретению, имеет важное значение для создания клеточных биомедицинских продуктов и генной терапии наследственных заболеваний человека.
Уровень техники
В настоящее время программируемые и специфичные нуклеазы являются основными инструментами геномной инженерии. В зависимости от их структуры выделяют 4 группы нуклеаз: мегануклеазы, нуклеазы цинковых пальцев (ZFN), эффекторные нуклеазы, подобные активаторам транскрипции (TALEN) и нуклеазы с кластеризованными регулярно расположенными короткими палиндромными повторами (CRISPR) [Gaj T., Gersbach C.A., Barbas C.F., 2013, ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering // Trends Biotechnol. 2013. Т. 31. № 7. С. 397-405]. Мегануклеазы, также известные как самонаводящиеся эндонуклеазы, представляют собой эндонуклеазы, характеризующиеся большим сайтом узнавания (14-40 п.н.) [Silva G., Poirot L., Galetto R. и др., 2011, Meganucleases and Other Tools for Targeted Genome Engineering: Perspectives and Challenges for Gene Therapy // Curr. Gene Ther. 2011. Т. 11. № 1. С. 11]. ZFN были созданы путем слияния ДНК-связывающих доменов цинковых пальцев белков с цинковыми пальцами с доменом расщепления эндонуклеазы FokI [Kim Y.G., Cha J., Chandrasegaran S., 1996, Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain // Proc. Natl. Acad. Sci. U.S.A. 1996. Т. 93. № 3. С. 1156-1160]. Специфичность последовательности ZFN обусловлена областью белка цинкового пальца, которая содержит от трех до шести “пальцев” Cys2-His2, каждый из которых распознает триплетный нуклеотидный код [Urnov F.D., Rebar E.J., Holmes M.C. и др., 2010, Genome editing with engineered zinc finger nucleases // Nat. Rev. Genet. 2010. Т. 11. № 9. С. 636-646]. Два белка «цинковых пальцев» связывают противоположные цепи ДНК в ближнем пространстве, позволяя слитой эндонуклеазе FokI образовывать функциональный димер, который расщепляет ДНК в целевых локусах [Smith J., Bibikova M., Whitby F.G. и др., 2000, Requirements for double-strand cleavage by chimeric restriction enzymes with zinc finger DNA-recognition domains // Nucleic Acids Res. 2000. Т. 28. № 17. С. 3361-3369]. Подобно ZFN, TALEN создаются путем слияния ДНК-связывающего домена, полученного из эффекторов, подобных активаторам транскрипции (TALE), и каталитического домена эндонуклеаз FokI [Miller J.C., Tan S., Qiao G. и др., 2011, A TALE nuclease architecture for efficient genome editing // Nat. Biotechnol. 2011. Т. 29. № 2. С. 143-148]. Системы CRISPR-Cas по существу представляют собой РНК-ориентированные нуклеазы. В отличие от вышеупомянутых нуклеаз, которые распознают целевую последовательность посредством взаимодействия белок-ДНК, нуклеазы CRISPR-Cas распознают целевые последовательности посредством спаривания оснований РНК и ДНК.
Открытие систем адаптивного иммунитета CRISPR-Cas у бактерий и архей [Makarova K.S., Wolf Y.I., Iranzo J. и др.,2020, Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants // Nat. Rev. Microbiol. 2020. Т. 18. № 2. С. 67-83] и демонстрация их применения для сайт-специфического редактирования генома бактерий и эукариот [Cong L., Ran F.A., Cox D. и др., 2013, Multiplex Genome Engineering Using CRISPR/Cas Systems // Science. 2013. Т. 339. № 6121. С. 819-823] дали толчок совершенно новой эре в синтетической биологии. За последнее десятилетие технология CRISPR-Cas приобрела широкую популярность в различных областях: от редактирования генома и обнаружения специфических последовательностей ДНК/РНК до контроля экспрессии генов. В основе этой технологии лежит возможность программирования комплексов CRISPR-Cas для воздействия на определенные локусы ДНК даже при использовании каталитически неактивных dCas-белков. Белки dCas9 можно использовать в качестве средств для адресной доставки связанных фрагментов в определенные локусы ДНК или для вмешательства в установленные взаимодействия белок-ДНК [Xu X., Qi L.S., 2019, A CRISPR-dCas Toolbox for Genetic Engineering and Synthetic Biology // J. Mol. Biol. 2019. Т. 431. № 1. С. 34-47]. Последняя идея привела к первому применению dCas9 для сайт-специфического контроля экспрессии генов посредством его вмешательства в связывание или продвижение РНК-полимеразы (РНКП) у прокариот (названное CRISPRi) [Qi L.S., Larson M.H., Gilbert L.A. и др., 2013, Repurposing CRISPR as an RNA-Guided Platform for Sequence-Specific Control of Gene Expression // Cell. 2013. Т. 152. № 5. С. 1173-1183]. Пул естественно полученных и сконструированных dCas-белков, включая слитые белки, представляет собой многообещающий набор инструментов, который можно использовать для создания функциональных синтетических генетических схем.
Метод редактирования генов на основе системы CRISPR/Cas имеет множество преимуществ перед ZFN и TALEN к которым относятся - простота разработки последовательностей направляющих РНК, компьютерное определение нецелевых эффектов на основе геномных последовательностей с высоким сходством с целевым локусом [Doench J.G., Fusi N., Sullender M. и др., 2016, Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9 // Nat. Biotechnol. 2016. Т. 34. № 2. С. 184-191] и возможность одновременного редактирования нескольких геномных локусов [Ho B.X., Loh S.J.H., Chan W.K. и др., 2018, In Vivo Genome Editing as a Therapeutic Approach // Int. J. Mol. Sci. 2018. Т. 19. № 9. С. 2721].
Чтобы направляемые нуклеазы могли иметь значение для генетического анализа и клинического применения, они должны демонстрировать высокую специфичность по отношению к целевым последовательностям ДНК. Однако сложные геномы часто содержат несколько копий последовательностей, которые идентичны или очень гомологичны целевой ДНК-мишени, что приводит к нецелевой активности и клеточной токсичности. Независимо от метода, концепция, лежащая в основе редактирования генов с помощью эндонуклеаз, заключается в индукции двухцепочечного разрыва (DSB) в целевых участках геномной ДНК клетки-хозяина, после чего репарация ДНК либо к случайным мутациям посредством негомологичного соединения концов (NHEJ), либо к целенаправленной мутации посредством гомологичной рекомбинации (HDR). Было показано, что путь HDR является наиболее доминирующим путем репарации DSB у дрожжей Saccharomyces cerevisiae по сравнению с путем NHEJ, тогда как у высших эукариотических видов верно обратное [Haber J.E., 2016, A Life Investigating Pathways That Repair Broken Chromosomes // Annu. Rev. Genet. 2016. Т. 50. С. 1-28]. Кроме того, путь репарации NHEJ классифицируется как подверженный ошибкам, поскольку ошибки в основном возникают при обработке концов разорванной двухцепочечной ДНК путем добавления или удаления нуклеотидов, что приводит к возникновению мутаций сдвига рамки считывания в транскрибируемых областях ДНК, которые могут быть либо мутагенными, либо смертельными для клеток [Chakraborty A., Tapryal N., Venkova T. и др., 2016, Classical non-homologous end-joining pathway utilizes nascent RNA for error-free double-strand break repair of transcribed genes // Nat. Commun. 2016. Т. 7. С. 13049]. В отличие от пути репарации NHEJ, путь HDR классифицируется как безошибочный, поскольку репарация DSB зависит не от добавления или удаления нуклеотидов на разорванных концах двухцепочечной ДНК, а от точного включения новых Фрагменты ДНК, гомологичные соседним последовательностям повреждений, основаны на гомологичной рекомбинации [Liu M., Rehman S., Tang X. и др., 2018, Methodologies for Improving HDR Efficiency // Front. Genet. 2018. Т. 9. С. 691]. Сообщалось и о токсичности инактивированных нуклеаз dCas9 в бактериальных клетках, что также объясняется неспецифическим связыванием белка с локусами геномной ДНК [Zhang S., Voigt C. A., 2018, Engineered dCas9 with reduced toxicity in bacteria: implications for genetic circuit design // Nucleic Acids Res. 2018. Т. 46. № 20. С. 11115-11125].
К настоящему времени существует несколько подходов к повышению специфичности и снижению нецелевой активности нуклеаз системы CRISPR/Cas. Один из способов - изменение аффинности Cas-белков путем введение мутаций (JP 2022141778). Предложенный Чжаном и Фойгтом подход состоит в том, чтобы уменьшить аффинность dCas путем мутации его домена, распознающего PAM, и одновременного слияния его с другим специфичным для последовательности транскрипционным фактором, который повышает как специфичность, так и аффинность всей конструкции. Другой подход - оптимизация дизайн направляющих РНК, например, показано уменьшение нецелевых эффектов при усечении комплементарной ДНК последовательности РНК на 1-3 нуклеотида [Wyvekens N., Topkar V.V., Khayter C. и др., 2015, Dimeric CRISPR RNA-Guided FokI-dCas9 Nucleases Directed by Truncated gRNAs for Highly Specific Genome Editing // Hum. Gene Ther. 2015. Т. 26. № 7. С. 425-431]. Недостатками обоих подходов является то, что сайт узнавания в 18-20 п.н. является довольно коротким и все остается вероятность его нахождения в другом месте генома, а также происходит снижение эффективности связывания.
Третий подход заключается в использовании парных никаз [Mali P., Aach J., Stranges P.B. и др., 2013, CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering // Nat. Biotechnol. 2013. Т. 31. № 9. С. 833-838]. Суть метода заключается в использовании никаз Cas9n с парой направляющих РНК, комплементарных противоположным цепям целевой последовательности, которые находятся в непосредственной близости. В то время как разрыв обеих цепей ДНК парой никаз Cas9 (D10A или H840A) приводит к сайт-специфическим DSB и NHEJ, отдельные разрывы преимущественно восстанавливаются с помощью высокоточного пути эксцизионного восстановления оснований (BER) [Dianov G.L., Hübscher U., 2013, Mammalian base excision repair: the forgotten archangel // Nucleic Acids Res. 2013. Т. 41. № 6. С. 3483-3490] или посредством гомологичной рекомбинации (HDR), поскольку приводят к генерации “липких” концов. Таким образом, происходит увеличение сайта узнавания в 2 раза, по сравнению с исходной системой, что снижает вероятность нецелевых взаимодействий. К тому же, одноцепочечные разрывы, которые могут образовываться в результате неспецифических взаимодействий будут приводить к одноцепочечным разрывам, которые гораздо легче подвергаются репарации. Используя подход парного разрыва, HDR становится более специфичным, чем нативный подход Cas9, с резким уменьшением количества нецелевых событий в 50-1500 раз без изменения эффективности расщепления на цели. Недостаток подхода заключается в том, что никазы не связаны между собой, что приводит к отсутствию кооперативности системы.
Последний подход заключается в использовании двух белков dCas9 слитых с нуклеазным доменом FokI [Aouida M., Eid A., Ali Z. и др., 2015, Efficient fdCas9 Synthetic Endonuclease with Improved Specificity for Precise Genome Engineering // PloS One. 2015. Т. 10. № 7. С. e0133373]. Идея состоит в том, что расщепление ДНК с помощью FokI-dCas9 (RFN) требует одновременного связывания двух различных мономеров FokI-dCas9, поскольку мономерные домены нуклеазы FokI не являются каталитически активными [Vanamee E.S., Santagata S., Aggarwal A.K., 2001, FokI requires two specific DNA sites for cleavage // J. Mol. Biol. 2001. Т. 309. № 1. С. 69-78]. В принципе, этот подход должен повысить специфичность расщепления ДНК, по сравнению с Cas9 дикого типа за счет удвоения количества указанных целевых оснований. Снижение токсичности обусловлено неактивностью мономера (RFN). Также следует отметить присутствие кооперативности в системе. Однако данный подход имеет и недостатки, среди которых - невозможность изменения расстояния между PAM-сайтами (только в диапазоне 15-25 п.н.), большой размер конструкции (может препятствовать эффективной доставке в клетки [Han H.A., Pang J.K.S., Soh B.-S., 2020, Mitigating off-target effects in CRISPR/Cas9-mediated in vivo gene editing // J. Mol. Med. Berl. Ger. 2020. Т. 98. № 5. С. 615-632]), сложность при добавлении дополнительных белковых эффекторных доменов, а также разрыв в 5-7 нуклеотидов (в случае парных никаз до 100 п.н.). Также сообщалось о том, что мономер FokI-dCas9, специфично связанный с ДНК, способен рекрутировать из второй другой мономер, не связанный с последовательностью, что потенциально приводит к нецелевым эффектам [Wyvekens N., Topkar V.V., Khayter C. и др., 2015, Dimeric CRISPR RNA-Guided FokI-dCas9 Nucleases Directed by Truncated gRNAs for Highly Specific Genome Editing // Hum. Gene Ther. 2015. Т. 26. № 7. С. 425-431].
Таким образом, техническая проблема, решаемая посредством заявляемого изобретения, заключается в необходимости преодоления недостатков, присущих аналогам и прототипу за счет разработки способа, позволяющего удобным образом повышать специфичность связывания CRISPR/dCas9 и CRISPR/dCas9 с таргетной последовательностью ДНК, а также придавать кооперативность системе. Как было описано ранее, решение такой проблемы актуально в связи с развитием технологий редактирования генома, эпигенома, создания и внедрения регуляторных генетических схем в клетки и организмы. Данный подход позволяет спроектировать системы, которые специфическим образом связываются с ДНК, при этом уровень эффективности их связывания оказывается выше, чем уровень специфичности связывания парных никаз без наличия димеризующихся доменов у молекул РНК.
Раскрытие изобретения
Техническим результатом заявляемого изобретения является разработка молекулярной системы специфическим образом связывающейся с двухцепочечными молекулами ДНК, при этом уровень эффективности ее связывания оказывается выше, чем уровень специфичности связывания отдельных Cas/РНК систем без наличия димеризующихся доменов у молекул РНК.
Технический результат достигается биомолекулярным комплексом для избирательного связывания участков двухцепочечной молекулы ДНК, включающим два Cas-белка и две молекулы РНК, каждая из которых состоит из трех фрагментов: гидовой РНК (гРНК), способной связывается с Cas-белком и целевой молекулой ДНК, 2) димеризующегося домена, способного связывать две молекулы РНК между собой и 3) линкерного фрагмента РНК, способного соединять гидовую РНК и димеризующийся домен. При этом построения комплекса выбирают такой тип взаимного расположения Cas-белков, при котором расстояние между кислородами, расположенными на 3' конце гРНК двух Cas-белков, будет наименьшим, при этом структуры Cas-белков и их гРНК стерически не взаимодействуют, а также отсутствуют стерические препятствия для построения РНК-фрагментов - 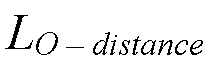 . Структуру димеризующегося домена РНК выбирают на основании его способности к димеризации со своей же копией, при этом выбирают димеризующийся домен РНК, длина которого
. Структуру димеризующегося домена РНК выбирают на основании его способности к димеризации со своей же копией, при этом выбирают димеризующийся домен РНК, длина которого 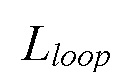 - не превышает половины
- не превышает половины 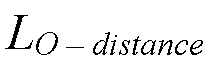 , затем для выбранного домена рассчитывают длину участка, участвующего в димеризации -
, затем для выбранного домена рассчитывают длину участка, участвующего в димеризации - 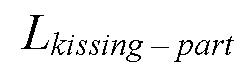 . Количество нуклеотидов, формирующих линкерный участок, рассчитывают по формуле (1):
. Количество нуклеотидов, формирующих линкерный участок, рассчитывают по формуле (1):
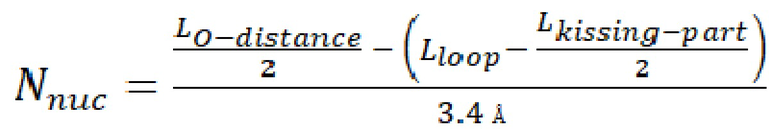 (1),
(1),
где 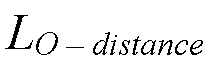 - расстояние между кислородами на 3'-концах гРНК Cas-белков,
- расстояние между кислородами на 3'-концах гРНК Cas-белков, 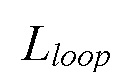 - длина выбранного димеризующегося РНК домена,
- длина выбранного димеризующегося РНК домена, 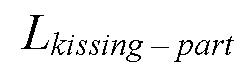 - длина участка, участвующего в kissing-loop взаимодействии, 3,4
- длина участка, участвующего в kissing-loop взаимодействии, 3,4 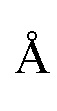 - константа, описывающая расстояние между парами азотистых оснований в составе РНК.
- константа, описывающая расстояние между парами азотистых оснований в составе РНК.
Нуклеотидный состав линкерного участка оптимизируют таким образом, чтобы линкерный участок не участвовал в формировании вторичных структур с димеризующимся РНК-фрагментом и участком гРНК.
Технический результат также достигается применением заявляемого ДНК-связывающего комплекса для модуляции экспрессии генов и создания генетических схем.
Также технический результат достигается применением заявляемого биомолекулярного комплекса (ДНК-связывающего) для создания высокоспецифичных систем редактирования генома.
Краткое описание чертежей
Изобретение поясняется следующим иллюстративным материалом.
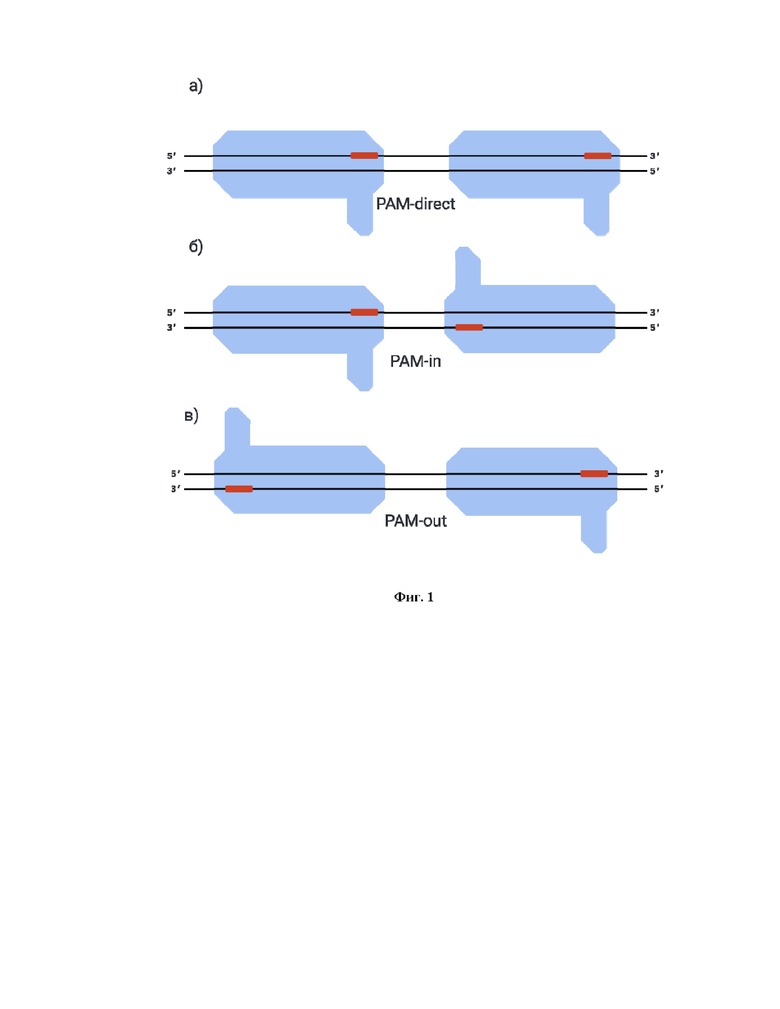
Фиг. 1 Способы взаимного расположения CAS-белков вдоль последовательности ДНК: а - PAM-direct, б - PAM-in, в - PAM-out.
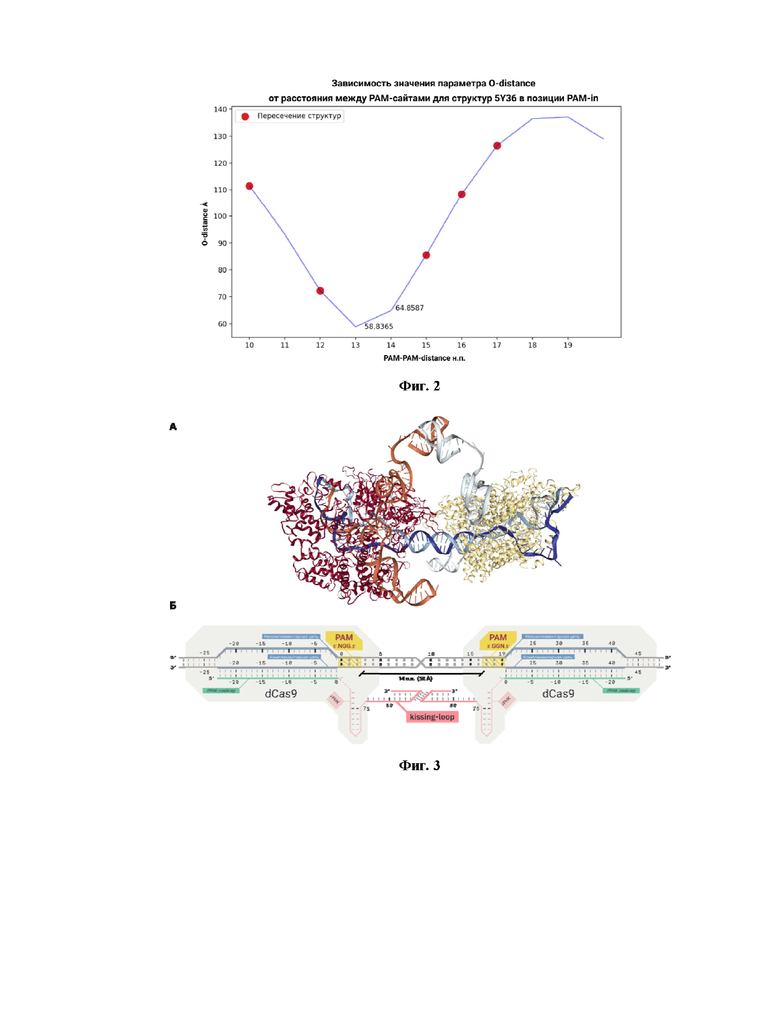
Фиг. 2 Зависимость значений расстояния между кислородами, расположенными на 3' конце гРНК двух Cas-белков (O-distance в 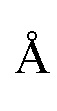 ), расположенных в положении PAM-in, от расстояния между PAM-сайтами (PAM-PAM-distance) в парах нуклеотидов. Пересечение структур - при данных значениях PAM-PAM-distance структуры 5Y36 стерически взаимодействуют.
), расположенных в положении PAM-in, от расстояния между PAM-сайтами (PAM-PAM-distance) в парах нуклеотидов. Пересечение структур - при данных значениях PAM-PAM-distance структуры 5Y36 стерически взаимодействуют.
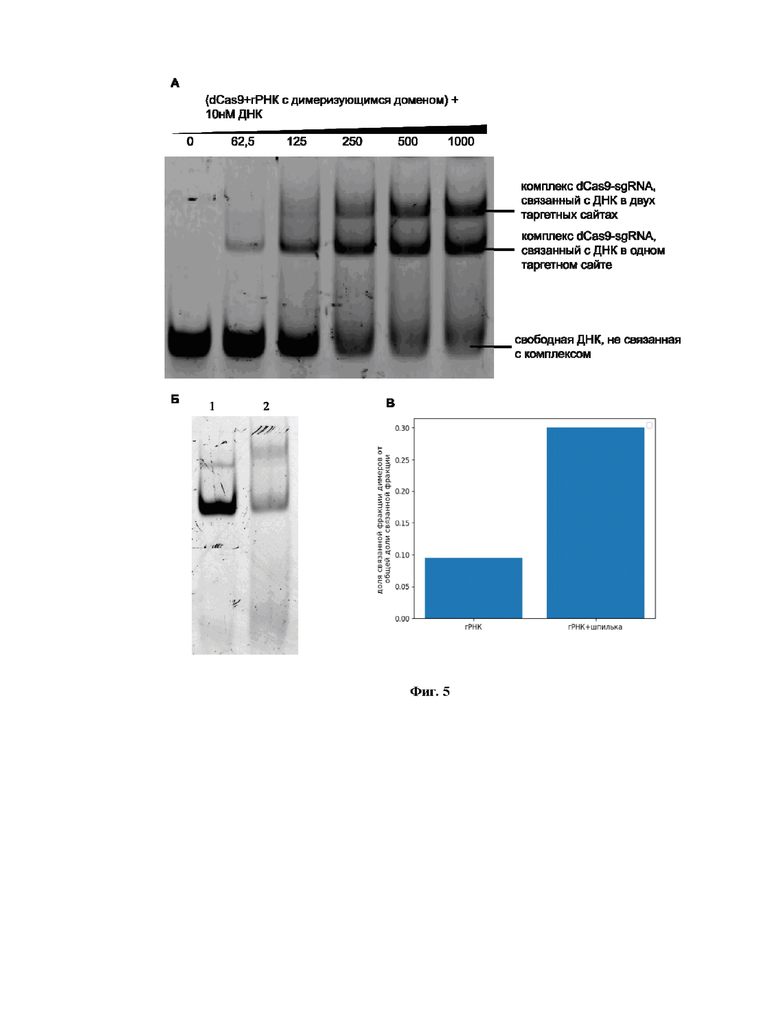
Фиг. 3 Пример молекулярной модели, состоящей из двух CRISPR-Cas систем, связанных со структурой двухцепочечной ДНК. В состав CRISPR-Cas систем входит гРНК в структуру которой добавлен димеризующийся РНК домен. А - результаты моделирования структуры бивалентной CRISPR/dCas9 системы, связанной димеризующимся РНК доменом, Б - принципиальная схема Cas-белвков в положении PAM-in, где PAM-PAM-distance 14 п.н.
Фиг. 4 Схема гидовой РНК молекулы с добавленным РНК-фрагментом, в состав которого входит линкерный участок и самодимеризующийся РНК домен. А - вторичная структура гидовой РНК с самодимеризующимся доменом. Б - последовательность гидовой РНК, где target - последовательность спейсерного участока гРНК, которая комплементарно связывается с таргетными сайтами в составе структуры двухцепочечной ДНК, gRNA scaffold - последовательность гРНК, отвечающая за пространственную конформацию гРНК, linker - линкерная последовательность в составе добавляемого РНК-фрагмента, loop - последовательность самодимеризующегося РНК домена в составе добавляемого РНК-фрагмента. В - Последовательность ДНК-матрицы для синтеза РНК с олигонуклеотидами, использованными для ее получения.
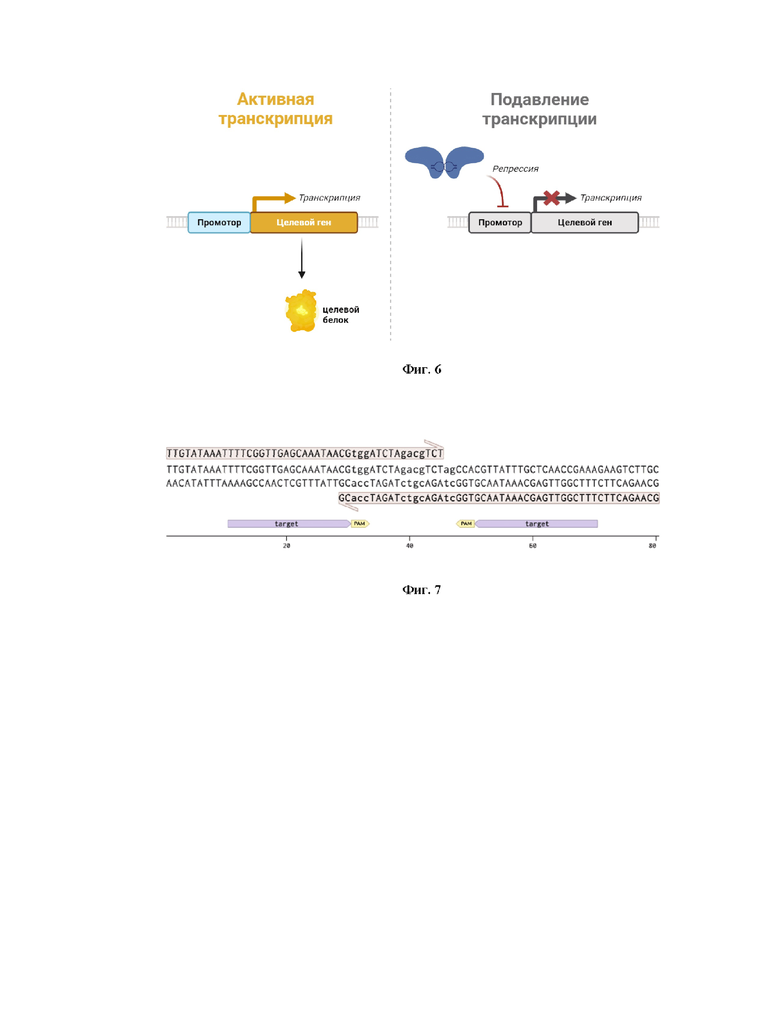
Фиг. 5 Проверка образования димерных комплексов dCas9-гРНК с двухцепочечной ДНК, где в состав гРНК входит димеризующийся домен. А - Анализ сдвига электрофоретической подвижности (EMSA) комплексов dCas9 с модифицированной гидовой РНК и ДНК, меченной 5'-6-FAM, доказывающий возможность образование комплексов, связанных через димеризующийся РНК-домен. Б - EMSA-анализ, отражающий сравнение эффективности сборки комплексов, связанных с двухцепочечной ДНК в двух таргетных сайтах при использовании гРНК с димеризующимся доменом, по сравнению с гРНК без димеризующегося домена, где 1 - комплекс dCas9-гРНК-ДНК без димеризующегося РНК-домена в последовательности гРНК, 2 - комплекс dCas9-гРНК-ДНК с димеризующимся РНК-доменом в последовательности гРНК. В - Диаграмма, показывающая сравнение доли образования димеров dCas9 от общей доли связанной фракции.
Фиг. 6 Схема использования в регуляторных генетических схемах.
Фиг. 7 Последовательность ДНК для связывания. target - таргетные сайты, PAM PAM-сайт.
Осуществление изобретения
Данное изобретение позволяет избирательно связывать систему, состоящую из Cas-белков с двухцепочечными молекулами ДНК для целей модуляции экспрессии генов или создания высокоспецифичных систем редактирования генома. Конкретная цель связывания определяется пользователем данного изобретения, как и последовательность ДНК с которой должна взаимодействовать система из Cas-белков. К примеру, для модуляции экспрессии флуоресцентного белка RFP в качестве сайта связывания для системы, состоящей из Cas-белков, будет выбрана последовательность ДНК, находящаяся непосредственно перед кодирующей последовательностью данного белка, в состав которой входит промоторный участок.
Подход основан на использовании двух Cas-белков и двух взаимодействующих молекул РНК входящих в состав комплекса CRISPR-Cas белков для создания ДНК-связывающего комплекса. В данном подходе молекулы РНК состоят из трех фрагментов: 1) гидовой РНК (гРНК), которая связывается с Cas-белком и позволяет ему избирательно связаться с участком двухцепочечной молекулы ДНК, 2) специального фрагмента - димеризующегося домена, который связывает две молекулы РНК между собой, 3) линкерного фрагмента РНК, соединяющего гидовую РНК и димеризующийся домен. Подробности конструирования системы раскрыты ниже.
Частью настоящего изобретения является способ на основе методов молекулярного моделирования, который на основе целевой последовательности ДНК, позволяет произвести дизайн молекул РНК таким образом, чтобы два Cas/РНК комплекса связались одновременно с ДНК и при этом между ними была установлена дополнительная связь посредством взаимодействия молекул РНК через димеризующийся домен.
При работе с протоколом используются специализированные программные библиотеки MDAnalysis, nglview для языка программирования Python, позволяющие модифицировать, визуализировать и проводить пространственный анализ файлов структур молекулярных систем, а также производить сборку систем, состоящих из нескольких отдельных структур. Альтернативой программных библиотек может служить программное обеспечение ChimeraX и PyMol, которое обладает схожим функционалом. Кроме этого используются веб-сервисы: nupack - сервис, предсказывающий вторичные структуры исходя из последовательности нуклеотидов РНК. Используется для оптимизации последовательности нуклеотидов, входящих в состав димеризующегося РНК домена, 3dRNA/DNA - предсказывает трехмерные структуры РНК исходя из последовательности нуклеотидов РНК. Используется для получения файла трехмерной структуры оптимизированной структуры РНК-фрагмента, x3DNA - для получения файлов трехмерных структур ДНК. В качестве альтернативы могут использоваться программы с графическим интересом, такие как: ChimeraX, PyMol, которые также можно использовать для получения структур двухцепочечной ДНК определенной последовательности нуклеотидов. Структуры CRISPR-Cas систем были взяты из банка данных “RCSB PDB”. В качестве модели dCas-белка нами была взята структура SpCas9 под идентификационным номером (PDB ID) 5Y36.
Частью настоящего изобретения является применение предлагаемых молекулярных систем для модуляции экспрессии генов и создания генетических схем. В этом случае в состав предлагаемых молекулярных комплексов входят каталитически неактивные производные Cas-белков - dCas-белки, а сами молекулярные комплексы используются для связывания ДНК в последовательностях генов и блокируют транскрипцию этих последовательностей, либо используются для доставки к промоторам генов транскрипционных и эпигенетических факторов, изменяющих экспрессию генов на эпигенетическом уровне.
Частью настоящего изобретения является применение предлагаемых молекулярных систем для создания высокоспецифичных систем редактирования генома. В этом случае в состав предлагаемых молекулярных комплексов входят производные Cas-белков, осуществляющие разрезание лишь одной цепи ДНК, - nCas-белки.
Подробное описание способа (протокола) конструирования молекулярных моделей предлагаемого класса систем, связывающихся с ДНК, позволяющего проводить дизайн необходимых последовательностей молекул РНК, входящих в состав комплекса
Способ может быть применен к различным семействам Cas-белков (Cas9, Cas12, Cas13, и другим семействам Cas-белков, обладающих свойством таргетного связывания с молекулой ДНК). Протокол состоит из нескольких шагов. Первый шаг - выбор взаимного расположения Cas-белков. В предлагаемом нами способе димеризующийся РНК домен используется для создания комплекса из двух Cas-белков, и выбор одного из вариантов взаимного позиционирования Cas-белков важен для дальнейшего расчета параметров димеризующегося РНК домена. При позиционировании двух Cas-белков вдоль последовательности ДНК существуют три варианта взаимного расположения Cas-белков: Pam-in, Pam-out, Pam-direct (Фиг. 1). При варианте расположения PAM-direct PAM-сайты двух Cas-белков расположены на одной цепи ДНК, таким образом, Cas-белки расположены последовательно друг за другом. При варианте расположения PAM-in и PAM-out PAM-сайты расположены на комплементарных цепях ДНК. В случае PAM-in PAM-сайты структурно сближены относительно PAM-out.
Шаг 1: Выбор взаимного расположения Cas-белков основан на анализе расстояний между кислородами, расположенными на 3' конце гРНК двух Cas-белков. При помощи программных библиотек или программного обеспечения описанного выше (MDAnalysis и nglview, или ChimeraX, PyMol), позволяющего модифицировать файлы структур, производится сборка структур, для каждого из вариантов взаимного расположения Cas-белков (PAM-in, PAM-out, PAM-direct), таким образом, чтобы Cas-белки максимально сближены, и стерически не препятствовали взаимодействию друг-друга со структурой двухцепочечной молекулой ДНК. Для оценки свободы стерических взаимодействий Cas-белков с ДНК оценивается значение минимального расстояния между структурами двух Cas-белков. Когда это расстояние принимает значения менее 3,5  , мы считаем, что Cas-белки начинают стерически взаимодействовать, где 3,5
, мы считаем, что Cas-белки начинают стерически взаимодействовать, где 3,5  это диаметр молекулы воды и значение, при котором принято считать, что структуры молекул стерически не конфликтуют. Если расстояние между структурами двух Cas-белков менее 3,5
это диаметр молекулы воды и значение, при котором принято считать, что структуры молекул стерически не конфликтуют. Если расстояние между структурами двух Cas-белков менее 3,5  их одновременное взаимодействие с двухцепочечной молекулой ДНК затруднительно. В случае, если участки структур Cas-белков, которые сближены более чем на 3,5
их одновременное взаимодействие с двухцепочечной молекулой ДНК затруднительно. В случае, если участки структур Cas-белков, которые сближены более чем на 3,5  , являются фрагментами белков, не участвующих во вторичной структуре белка (например, “петли”), то необходимо учитывать возможную подвижность данных участков при анализе структур. Также необходимо учитывать последовательности PAM-сайтов при моделировании Cas-белков в комплексе в ДНК. Процесс сборки структур из двух Cas-белков происходит путем пространственного выравнивания ДНК нуклеотидов, входящих в структуру CRISPR-Cas систем на структуру линейной ДНК и дальнейшего их объединения. Структура линейной ДНК может быть получена при помощи веб-сервиса x3DNA, или смоделирована в программе ChimeraX. После сборки структур производится сравнение полученных систем путем расчета расстояний между кислородами, расположенными на 3' конце гРНК двух Cas-белков (
, являются фрагментами белков, не участвующих во вторичной структуре белка (например, “петли”), то необходимо учитывать возможную подвижность данных участков при анализе структур. Также необходимо учитывать последовательности PAM-сайтов при моделировании Cas-белков в комплексе в ДНК. Процесс сборки структур из двух Cas-белков происходит путем пространственного выравнивания ДНК нуклеотидов, входящих в структуру CRISPR-Cas систем на структуру линейной ДНК и дальнейшего их объединения. Структура линейной ДНК может быть получена при помощи веб-сервиса x3DNA, или смоделирована в программе ChimeraX. После сборки структур производится сравнение полученных систем путем расчета расстояний между кислородами, расположенными на 3' конце гРНК двух Cas-белков ( 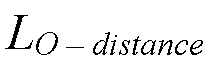 ). Именно к этим атомам кислорода в дальнейшем будет добавляться РНК-фрагмент, в составе которого будет димеризующийся РНК домен. Выбирается тот вариант взаимного позиционирования Cas-белков, в котором расстояние между кислородами на 3'-концах гРНК наименьшее, а также между выбранными кислородами отсутствуют стерические препятствия для построения РНК-фрагментов. Под стерическим препятствием для построения РНК-фрагментов могут подразумеваться структуры белка или РНК попадающие в зону предполагаемого расположения будущего РНК-фрагмента. Зона описывается как цилиндр с основаниями в координатах расположения кислородов на 3'-концах гРНК и диаметром целина порядка 20-30
). Именно к этим атомам кислорода в дальнейшем будет добавляться РНК-фрагмент, в составе которого будет димеризующийся РНК домен. Выбирается тот вариант взаимного позиционирования Cas-белков, в котором расстояние между кислородами на 3'-концах гРНК наименьшее, а также между выбранными кислородами отсутствуют стерические препятствия для построения РНК-фрагментов. Под стерическим препятствием для построения РНК-фрагментов могут подразумеваться структуры белка или РНК попадающие в зону предполагаемого расположения будущего РНК-фрагмента. Зона описывается как цилиндр с основаниями в координатах расположения кислородов на 3'-концах гРНК и диаметром целина порядка 20-30  , что соответствует средним значениям ширины двухцепочечной РНК. Кроме этого стоит учитывать способ взаимодействия Cas-белка выбранного семейства со структурой двухцепочечной молекулы ДНК [Wang J.Y., Pausch P., Doudna J.A., 2022, Structural biology of CRISPR-Cas immunity and genome editing enzymes // Nat. Rev. Microbiol. 2022. Т. 20. № 11. С. 641-656].
, что соответствует средним значениям ширины двухцепочечной РНК. Кроме этого стоит учитывать способ взаимодействия Cas-белка выбранного семейства со структурой двухцепочечной молекулы ДНК [Wang J.Y., Pausch P., Doudna J.A., 2022, Structural biology of CRISPR-Cas immunity and genome editing enzymes // Nat. Rev. Microbiol. 2022. Т. 20. № 11. С. 641-656].
Шаг 2: После выбора типа взаимного расположения Cas-белков необходимо рассчитать, на каком расстоянии будут находиться кислороды на 3'-концах гРНК выбранных Cas-белков в заданном расположении. Расстояние между кислородами на 3'-концах гРНК Cas-белков называем 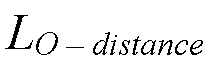 . Результатом второго шага является структура комплекса состоящего из двух Cas-белков взаимодействующих с двухцепочечной ДНК и значение параметра
. Результатом второго шага является структура комплекса состоящего из двух Cas-белков взаимодействующих с двухцепочечной ДНК и значение параметра 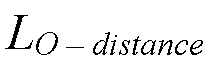 , рассчитанного для этой структуры.
, рассчитанного для этой структуры.
Шаг 3: Подготовка и анализ вводимого РНК-фрагмента, в состав которого входит димеризующийся РНК домен. Выбор структуры димеризующегося РНК домена основывается на его способности к димеризации со своей же копией, так называемые kissing-loop взаимодействия [Liu D., Geary C.W., Chen G. и др., 2020, Branched kissing loops for the construction of diverse RNA homooligomeric nanostructures // Nat. Chem. 2020. Т. 12. № 3. С. 249-259]. Структура димеризующегося РНК домена может быть взята из банка данных RCSB PDB (пример 2D1B). Или сгенерирована при помощи веб-сервисов: nupack - для оптимизации последовательности нуклеотидов, входящих в состав димеризующегося РНК домена, 3dRNA/DNA - для получения файла трехмерной структуры, оптимизированного димеризующегося РНК домена. Для файла структуры димеризующегося РНК домена рассчитывается его длина - 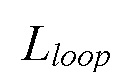 . Длина структуры димеризующегося РНК домена не должна превышать половины
. Длина структуры димеризующегося РНК домена не должна превышать половины 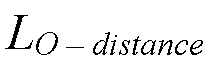 . Для соответствия данным условиям выбранный димеризующийся РНК домен может быть отредактирован с сохранением участка, участвующего непосредственно в kissing-loop взаимодействиях.
. Для соответствия данным условиям выбранный димеризующийся РНК домен может быть отредактирован с сохранением участка, участвующего непосредственно в kissing-loop взаимодействиях.
Шаг 4: Для выбранной структуры димеризующегося РНК домена производится расчет длины участка, участвующего непосредственно в димеризации РНК доменов ( 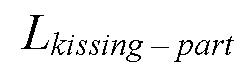 ). Расчет производится при анализе файла структуры димеризующегося РНК домена, полученного на предыдущем шаге. Расчет длины
). Расчет производится при анализе файла структуры димеризующегося РНК домена, полученного на предыдущем шаге. Расчет длины 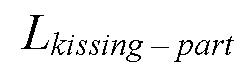 проводится при помощи программных библиотек или программного обеспечения описанного выше (MDAnalysis и nglview, или ChimeraX, PyMol) позволяющего работать с pdb-файлами. Измеряется максимальное расстояние между атомами нуклеотидов участвующих в димеризации РНК доменов.
проводится при помощи программных библиотек или программного обеспечения описанного выше (MDAnalysis и nglview, или ChimeraX, PyMol) позволяющего работать с pdb-файлами. Измеряется максимальное расстояние между атомами нуклеотидов участвующих в димеризации РНК доменов.
Шаг 5: Результатом 5 шага является структура РНК-фрагмента состоящего из линкерного участка и димеризующегося РНК домена. Для этого производится расчет количества нуклеотидов, входящих в состав линкерного участка, необходимого для увеличения гибкости и длины добавляемого фрагмента. Для этого используется формула 1:
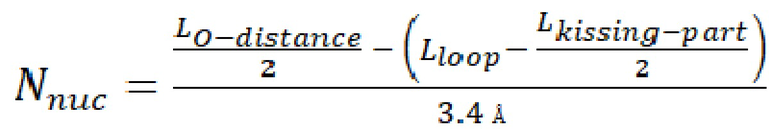 (1)
(1)
где 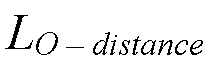 - расстояние между кслородами на 3'-концах гРНК Cas-белков,
- расстояние между кслородами на 3'-концах гРНК Cas-белков, 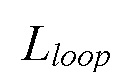 - длина выбранного димеризующегося РНК домена,
- длина выбранного димеризующегося РНК домена, 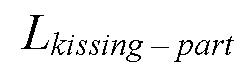 - длина участка, участвующего в kissing-loop взаимодействии, 3,4
- длина участка, участвующего в kissing-loop взаимодействии, 3,4  - константа описывающая расстояние между парами азотистых оснований в составе РНК.
- константа описывающая расстояние между парами азотистых оснований в составе РНК.
Нуклеотидный состав линкерного участка оптимизируется при помощи веб-сервиса Nupack, способного предсказывать вторичные структуры РНК таким образом, чтобы линкерный участок не участвовал в формировании вторичных структур с димеризующимся РНК-фрагментом и участком гРНК в последовательность которого он будет вводиться.
Файл структуры РНК-фрагмента состоящего из линкерной последовательности и димеризующегося РНК домена, полученного на 3 шаге, создается при помощи программного обеспечения PyMol или веб-сервиса 3dRNA/DNA. В функционал данных инструментов входит создание файлов структур РНК с заданной последовательностью.
Шаг 6: Сборка молекулярной модели комплекса, полученного на втором шаге с добавлением структуры РНК-фрагмента в состав гРНК Cas-белков, полученной на пятом шаге. Для этого структура гРНК каждого из двух Cas-белков соединяться со структурой РНК-фрагмента. Координаты кислорода в составе фосфатной группы, расположенного на 5'-конце РНК-фрагмента принимают значения координат кислорода, расположенного на 3'-конце гРНК. При этом структурам РНК-фрагментов задается направление в сторону противоположного Cas-белка (Фиг. 3). Весь процесс модификации и визуализации проводится с использованием программных библиотек MDAnalysis и nglview для языка программирования Python, они позволяют модифицировать, визуализировать и проводить пространственный анализ файлов структур молекулярных систем, а также производить сборку систем, состоящих из нескольких отдельных структур.
Итогом применения данного протокола является молекулярная модель, которая теоретически описывает способ взаимодействия системы, состоящей из Cas-белков с двухцепочечной молекулой ДНК. Данный протокол может быть применен несколько раз с целью сборки молекулярных моделей, различающихся по расстоянию между Cas-белками, по варианту взаимной ориентации Cas-белков в системе, и по структуре димеризующегося РНК домена. Модели, полученные в результате выполнения протокола, могут быть сравнены между собой при помощи программных библиотек MDAnalysis и nglview, или программного обеспечения ChimeraX и PyMol.
Подробнее способ конструирования ДНК-связывающего комплекса и его экспериментальная проверка проиллюстрированы в виде примера ниже.
В приведенном ниже примере использования протокола нами преследовалась цель по подтверждению образования и сборки систем, состоящих из двух Cas-белков. Для достижения поставленной цели была выбрана последовательность двухцепочечной ДНК (см. раздел “последовательности”). На основе последовательности ДНК с таргетными сайтами был изменен спейсерный участок последовательности гРНК с целью комплементарного взаимодействия гРНК в составе комплекса Cas-белка с таргетными сайтами в составе структуры двухцепочечной ДНК (Фиг. 4 Б).
На основе представленного выше протокола была собрана система на основе белка SpCas9, в качестве структуры которого была взята структура под идентификационным номером (PDB ID) 5Y36 из банка данных RCSB PDB. Процесс сборки и анализа получаемых структур проводился при помощи программной библиотеки MDAnalysis для языка программирования Python версии 3.10.
Шаг 1: Выбор взаимного расположения Cas-белков основан на анализе расстояний между кислородами, расположенными на 3' конце гРНК двух Cas-белков. При помощи программной библиотеки MDAnalysis позволяющей модифицировать файлы структур молекул (pdb-file), была произведена сборка структур, для каждого из вариантов взаимного расположения Cas-белков (PAM-in, PAM-out, PAM-direct), таким образом, что Cas-белки максимально сближены, и стерически не препятствуют взаимодействию друг-друга со структурой двухцепочечной молекулой ДНК. Параметры, по которым определяется стерическое взаимодействие, подробно описаны в соответствующем пункте протокола. Для описания структур, полученных в данном примере, использовали характеристику PAM-PAM-distance - расстояние в нуклеотидах между PAM-сайтами двух Cas-белков. Стоит отметить, что параметр PAM-PAM-distance не описывает, насколько структуры Cas-белков сближены, а лишь указывает количество нуклеотидов между PAM-сайтами. Расстояние между кислородами на 3' концах гРНК обозначено как O-distance. Так для PAM-in минимальное значение параметра PAM-PAM-distance составило 13 п.н., а расстояние O-distance составило порядка 60  , для PAM-out минимальное значение параметра PAM-PAM-distance составило 51 п.н., а расстояние O-distance составило порядка 30
, для PAM-out минимальное значение параметра PAM-PAM-distance составило 51 п.н., а расстояние O-distance составило порядка 30  , для PAM-direct минимальное значение параметра PAM-PAM-distance составило 37 п.н., а расстояние O-distance составило порядка 65
, для PAM-direct минимальное значение параметра PAM-PAM-distance составило 37 п.н., а расстояние O-distance составило порядка 65  . Для дальнейшей работы было решено выбрать тип взаимной ориентации PAM-in. Согласно разработанному протоколу выбирается тот вариант взаимного позиционирования Cas-белков, в котором расстояние между кислородами на 3'-концах гРНК наименьшее, а также между выбранными кислородами отсутствуют стерические препятствия для построения РНК-фрагментов. Кроме этого, стоит учитывать способ взаимодействия Cas-белка выбранного семейства со структурой двухцепочечной молекулы ДНК [Wang J.Y., Pausch P., Doudna J. A., 2022, Structural biology of CRISPR-Cas immunity and genome editing enzymes // Nat. Rev. Microbiol. 2022. Т. 20. С. 641-656]. Для каждого из способов взаимной ориентации Cas-белков рассчитывалась зона предполагаемого расположения будущего РНК-фрагмента. Согласно расчетам был выбран тип взаимного расположения Cas-белков PAM-in, поскольку в нем наблюдалось наименьшее количество стерических препятствий для построения РНК-фрагментов. Однако стоит отметить, что вариант расположения Cas-белков PAM-out также является возможным.
. Для дальнейшей работы было решено выбрать тип взаимной ориентации PAM-in. Согласно разработанному протоколу выбирается тот вариант взаимного позиционирования Cas-белков, в котором расстояние между кислородами на 3'-концах гРНК наименьшее, а также между выбранными кислородами отсутствуют стерические препятствия для построения РНК-фрагментов. Кроме этого, стоит учитывать способ взаимодействия Cas-белка выбранного семейства со структурой двухцепочечной молекулы ДНК [Wang J.Y., Pausch P., Doudna J. A., 2022, Structural biology of CRISPR-Cas immunity and genome editing enzymes // Nat. Rev. Microbiol. 2022. Т. 20. С. 641-656]. Для каждого из способов взаимной ориентации Cas-белков рассчитывалась зона предполагаемого расположения будущего РНК-фрагмента. Согласно расчетам был выбран тип взаимного расположения Cas-белков PAM-in, поскольку в нем наблюдалось наименьшее количество стерических препятствий для построения РНК-фрагментов. Однако стоит отметить, что вариант расположения Cas-белков PAM-out также является возможным.
Шаг 2: Для выбранного типа взаимного расположения Cas-белков, в нашем случае PAM-in, мы произвели расчет расстояния между кислородами на 3'-концах гРНК структуры 5Y36 в выбранном расположении. Расстояние между кислородами на 3'-концах гРНК Cas-белков называем 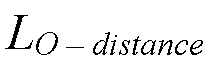 . Результат анализа представлен в кратком описании чертежей (Фиг. 2), по нему можно сделать вывод, что структуры с PAM-PAM-distance равным 13 и 14 п.н. являются наиболее перспективными. В то время как структура со значением PAM-PAM-distance равным 11 п.н. имеет большее расстояние между кислородами чем структуры со значениями в 13 и 14 п.н.. Также структуры Cas-белков в системе со значением PAM-PAM-distance равным 13 п.н. более сближены чем в системе со значением PAM-PAM-distance равным 14 п.н. Исходя из вышеописанных параметров систем была выбрана система с PAM-PAM-distance равным 14 п.н, для уменьшения вероятности возможных стерических взаимодействий между Cas-белками во время их взаимодействий со структурой двухцепочечной ДНК. Значение
. Результат анализа представлен в кратком описании чертежей (Фиг. 2), по нему можно сделать вывод, что структуры с PAM-PAM-distance равным 13 и 14 п.н. являются наиболее перспективными. В то время как структура со значением PAM-PAM-distance равным 11 п.н. имеет большее расстояние между кислородами чем структуры со значениями в 13 и 14 п.н.. Также структуры Cas-белков в системе со значением PAM-PAM-distance равным 13 п.н. более сближены чем в системе со значением PAM-PAM-distance равным 14 п.н. Исходя из вышеописанных параметров систем была выбрана система с PAM-PAM-distance равным 14 п.н, для уменьшения вероятности возможных стерических взаимодействий между Cas-белками во время их взаимодействий со структурой двухцепочечной ДНК. Значение 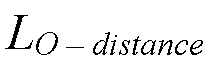 составило порядка 65
составило порядка 65  .
.
Шаг 3: Для файла структуры димеризующегося РНК домена рассчитывается его длина - 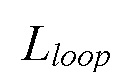 . Длина структуры димеризующегося РНК домена не должна превышать половины
. Длина структуры димеризующегося РНК домена не должна превышать половины 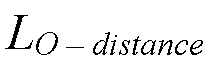 . Согласно предыдущим шагам протокола
. Согласно предыдущим шагам протокола 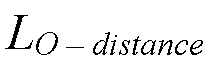 ≈ 65
≈ 65  , соответственно, длина димеризующегося РНК домена не должны превышать 32,5
, соответственно, длина димеризующегося РНК домена не должны превышать 32,5  (
( 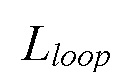 ≤ 32,5
≤ 32,5  ). Для соответствия данным условиям выбранный димеризующийся РНК домен может быть отредактирован с сохранением участка, участвующего непосредственно в kissing-loop взаимодействиях. В нашем случае была взята структура димеризующегося РНК домена из банка данных RCSB PDB, PDB ID: 2D1B. Структура 2D1B изначально имела длину порядка 55
). Для соответствия данным условиям выбранный димеризующийся РНК домен может быть отредактирован с сохранением участка, участвующего непосредственно в kissing-loop взаимодействиях. В нашем случае была взята структура димеризующегося РНК домена из банка данных RCSB PDB, PDB ID: 2D1B. Структура 2D1B изначально имела длину порядка 55  , что превышало возможные значения дины димеризующегося РНК домена (
, что превышало возможные значения дины димеризующегося РНК домена (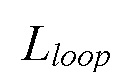 ≤ 32,5
≤ 32,5  ). Для соответствия этому параметру структуры 2D1B была отредактирована. Во время редактирования при помощи программной библиотеки MDAnalysis из структуры 2D1B удалялись комплиментарные пары нуклеотидов с 3' и 5' концов РНК, не участвующего непосредственно в kissing-loop взаимодействиях. Отредактированная структура 2D1B имела длину
). Для соответствия этому параметру структуры 2D1B была отредактирована. Во время редактирования при помощи программной библиотеки MDAnalysis из структуры 2D1B удалялись комплиментарные пары нуклеотидов с 3' и 5' концов РНК, не участвующего непосредственно в kissing-loop взаимодействиях. Отредактированная структура 2D1B имела длину 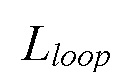 = 30
= 30  . Последовательность отредактированной структуры 2D1B указана ниже (см. последовательности).
. Последовательность отредактированной структуры 2D1B указана ниже (см. последовательности).
Шаг 4: Для полученной структуры димеризующегося РНК домена на предыдущем шаге производится расчет длины участка, участвующего непосредственно в димеризации РНК доменов ( 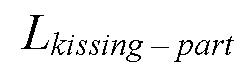 ). Расчет значений
). Расчет значений 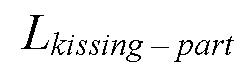 проводился при помощи программной библиотеки MDAnalysis, где рассчитывается расстояние между наиболее удаленными нуклеотидами учавствующими в kissing-loop взаимодействии. Длина участка, участвующего непосредственно в kissing-loop составила 20
проводился при помощи программной библиотеки MDAnalysis, где рассчитывается расстояние между наиболее удаленными нуклеотидами учавствующими в kissing-loop взаимодействии. Длина участка, участвующего непосредственно в kissing-loop составила 20  .
.
Шаг 5: Согласно формуле 1 был произведен расчет количества нуклеотидов, входящих в состав линкерного участка. Данный участок необходим для увеличения гибкости и длины добавляемого фрагмента. Пример расчета по формуле 1:
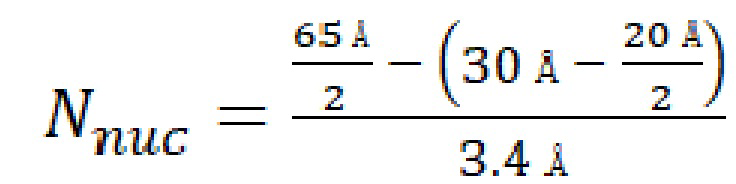 (1)
(1)
где 65  - расстояние между кислородами на 3'-концах гРНК Cas-белков, расстояние между кислородами на 3'-концах гРНК Cas-белков, 30
- расстояние между кислородами на 3'-концах гРНК Cas-белков, расстояние между кислородами на 3'-концах гРНК Cas-белков, 30  - длина выбранного димеризующегося РНК домена, 20
- длина выбранного димеризующегося РНК домена, 20  - длина участка, участвующего в kissing-loop взаимодействии, 3,4
- длина участка, участвующего в kissing-loop взаимодействии, 3,4  - константа описывающая расстояние между парами азотистых оснований в составе РНК.
- константа описывающая расстояние между парами азотистых оснований в составе РНК.
Количество нуклеотидов линкерного участка (Nnuc) составило 4 нуклеотида. Нуклеотидный состав линкерного участка оптимизируется при помощи веб-сервиса Nupack, способного предсказывать вторичные структуры РНК таким образом, чтобы линкерный участок не участвовал в формировании вторичных структур с димеризующимся РНК-фрагментом и участком гРНК в последовательность которого он будет вводиться.
Файл структуры РНК-фрагмента состоящего из линкерной последовательности и димеризующегося РНК домена, полученного на третьем шаге, создается при помощи программного обеспечения PyMol или веб-сервиса 3dRNA/DNA. В функционал данных инструментов входит создание файлов структур РНК с заданной последовательностью. Результатом пятого шага является структура РНК-фрагмента, которая в дальнейшем будет добавлен к структуре гРНК Cas-белков.
Шаг 6: Сборка молекулярной модели комплекса, полученного на втором шаге с добавлением структуры РНК-фрагмента в состав гРНК Cas-белков, полученной на пятом шаге. Для этого структура гРНК каждого из двух Cas-белков соединяться со структурой РНК-фрагмента. Координаты кислорода в составе фосфатной группы, расположенного на 5'-конце РНК-фрагмента принимают значения координат кислорода, расположенного на 3'-конце гРНК. При этом структурам РНК-фрагментов задается направление в сторону противоположного Cas-белка (Фиг. 3). Весь процесс модификации и визуализации проводится с использованием программных библиотек MDAnalysis и nglview для языка программирования Python, они позволяют модифицировать, визуализировать и проводить пространственный анализ файлов структур молекулярных систем, а также производить сборку систем, состоящих из нескольких отдельных структур.
Последовательности:
Двухцепочечная ДНК:
5'-TTGTATAAATTTTCGGTTGAGCAAATAACGTGGATCTAGACGTCTAGCCACGTTATTTG CTCAACCGAAAGAAGTCTTGC-3'
Последовательность ДНК для связывания с комплексом dCas9-гРНК, представлена на Фиг. 7.
гРНК: GGUACCGCUCCAGUCGUUCAUGGUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGGCUAG UCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCU
Линкерный участок:
GUGA
Димеризующийся РНК домен:
UGCUGAAGUGCACACGGCA
Собранная на основе разработанного протокола модель системы и ее схема приведены на Фиг. 3, а последовательность молекулы РНК и образовывающие ее вторичные структуры на Фиг. 4.
Очистка белка dCas9
Для экспрессии белка dCas9 использовали плазмиду pMJ841, кодирующую белок dCas9 со слитым на N-конце белком MBP и полигистидиновым тагом (6xHis) для очистки. Экспрессию проводили в клетках BL21(DE3). Дальнейшую очистку проводили в три этапа с использованием хроматографической системы ÄKTA FPLC system (GE Healthcare). На первом этапе проводили металл-аффинную хроматографию с использованием никелевой колонки HiTrap Chelating HP (Cytiva). Белок dCas9, слитый с 6xHis-MBP, элюировали с использованием линейного градиента имидазола. Далее проводили протеолиз для удаления конструкции 6xHis-MBP. Очищенный белок на первом этапе белок диализовали и очищали с использованием катионообменной хроматографии на колонке HiTrap SP FF. Для улучшения степени очистки проводили гель-фильтрацию с использованием колонки Superose 6 Increase 10/300 GL. Фракции, содержащие белок, концентрировали и до использования хранили на -80°C.
Синтез и подбор олигонуклеотидов
Для проведения экспериментальных исследований были использованы олигонуклеотиды, приготовленные по заказу специализированной биотехнологической фирмой. Синтезируют (ЗАО «Евроген», Россия; по заказу Биологического факультета МГУ им. М. В. Ломоносова) набор олигонуклеотидов для наработки последовательностей ДНК-матрицы для транскрипции, а также ДНК-последовательностей для связывания с комплексом dCas9-гРНК. В случае ДНК-матрицы для транскрипции использовали набор из четырех частично комплементарных олигонуклеотидов, образующих перекрытия в трех областях - промоторной (с 1 по 19 нуклеотид), а также в области начала (с 44 по 59 нуклеотид) и конца (с 103 по 116 нуклеотид) последовательности гРНК, отвечающей за пространственную конформацию (Фиг. 4 В). Области перекрытия выбирали таким образом, чтобы разницы температуры отжига всех областей составляли не более 10°C. Подбор праймеров проводили с использованием платформы Benchling (https://www.benchling.com/).
В случае ДНК-последовательностей для связывания с комплексом dCas9-гРНК олигонуклеотиды синтезированы так, что перекрытие находится в области 29-45 нуклеотидов (Фиг. 7). Прямой праймер заказывали с флуоресцентной меткой 6-FAM на 5'-конце.
Необходимые для наработки ДНК и РНК последовательностей реактивы заказывали в компании New England Biolabs (США).
Получение ДНК и РНК-последовательностей
Направляющие РНК синтезировали методом in vitro транскрипции с использованием T7 РНК-полимеразы. ДНК-матрицу для транскрипции нарабатывали путем отжига частично комплементарных олигонуклеотидов с последующим удлинением концов высокоточной полимеразой Q5. ДНК-последовательности для связывания также получали путем отжига олигонуклеотидов с частичной комплементарностью и последующим удлинением концов высокоточной Q5-полимеразой.
Сборка комплексов dCas9-гРНК и dCas9-гРНК-ДНК
Для сборки тройного комплекса dCas9-гРНК-ДНК сначала собирали рибонуклеопротеидный комплекс, состоящий из dCas9-гРНК. Для этого в пробирку с реакционным буфером добавляли белок и двукратный избыток гРНК и инкубировали при 37°C. После часовой инкубации к комплексу добавляли очищенную ДНК, меченную на 5'-конце флуоресцентным красителем 6-FAM. Инкубацию с ДНК также проводили при 37°C в течение 1 часа.
Система была протестирована экспериментально в системе in vitro (Фиг. 5). Методом анализа сдвига электрофоретической подвижности было подтверждено наличие комплексов, состоящих из димеров dCas9-белков, связанных РНК-доменом (Фиг. 5 А). На Фиг. 5 А видно, что с увеличением концентрации комплекса dCas9-гРНК увеличивается доля dCas9-белков, связанных через димеризующийся РНК-домен с двумя таргетными сайтами на двухцепочечной ДНК. Так, при концентрации dCas9-гРНК в 1мкМ порядка половины комплексов dCas9-гРНК связываются с ДНК в двух таргетных сайтах. При низкой концентрации dCas9-гРНК образуются комплексы, связанные с двухцепочечной ДНК в одном таргетном сайте. Поскольку комплексы dCas9-гРНК можно направить на два расположенных рядом таргетных сайта и без димеризующегося домена (подход парных никаз), мы сравнили эффективность связывания комплексов dCas9-гРНК с двумя таргетными сайтами при наличии димеризующихся РНК-доменов в составе гРНК и при их отсутствии в составе гРНК. Используя метод анализа сдвига электрофоретической подвижности и последующий анализ данных в программе ImageJ, нами показано, что наличие димеризующегося РНК-домена в последовательности гРНК приводит к кооперативным эффектам, что в 3 раза повышает вероятность образования комплексов, связанных с двумя таргетными сайтами по сравнению с гРНК без димеризующегося РНК домена (Фиг. 5 Б, В).
| название | год | авторы | номер документа |
|---|---|---|---|
| ОПОСРЕДОВАННАЯ НУКЛЕАЗОЙ СБОРКА ДНК | 2015 |
|
RU2707911C2 |
| СИСТЕМА РЕДАКТИРОВАНИЯ ГЕНОМНОЙ ДНК ЭУКАРИОТИЧЕСКОЙ КЛЕТКИ НА ОСНОВЕ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ, КОДИРУЮЩЕЙ БЕЛОК SUCAS9NLS | 2022 |
|
RU2804422C1 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ НАЦЕЛЕННОЙ ГЕНЕТИЧЕСКОЙ МОДИФИКАЦИИ С ИСПОЛЬЗОВАНИЕМ ПАРНЫХ ГИДОВЫХ РНК | 2015 |
|
RU2734770C2 |
| ИСКУССТВЕННО СОЗДАННАЯ СИСТЕМА УПРАВЛЕНИЯ ФУНКЦИЕЙ ШК | 2017 |
|
RU2768043C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МОДИФИКАЦИИ ЦЕЛЕВОГО ЛОКУСА | 2015 |
|
RU2704283C2 |
| Средство разрезания двунитевой ДНК с помощью Cas12d белка из Katanobacteria и гибридной РНК, полученной путем слияния направляющей CRISPR РНК и scout РНК | 2020 |
|
RU2771626C1 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ПОЛУЧЕНИЯ ИЛИ ПОДДЕРЖАНИЯ ПЛЮРИПОТЕНТНЫХ КЛЕТОК | 2015 |
|
RU2706965C2 |
| Средство разрезания ДНК на основе Cas9 белка из бактерии Streptococcus uberis NCTC3858 | 2022 |
|
RU2788197C1 |
| ОРТОГОНАЛЬНЫЕ БЕЛКИ CAS9 ДЛЯ РНК-НАПРАВЛЯЕМОЙ РЕГУЛЯЦИИ И РЕДАКТИРОВАНИЯ ГЕНОВ | 2021 |
|
RU2771583C1 |
| НАПРАВЛЯЕМАЯ РНК РЕГУЛЯЦИЯ ТРАНСКРИПЦИИ | 2014 |
|
RU2690935C2 |

Настоящее изобретение относится к области биотехнологии, в частности к системам управления экспрессией генов, системам создания синтетических генетических схем в живых клетках, системам адресной доставки молекул к областям генома, системам редактирования генома и эпигенома на основе нуклеотидной последовательности, кодирующей белки nCas9/dCas9, и нуклеотидных последовательностей, кодирующих молекулы РНК с самодимеризующимися РНК доменами, в качестве инструмента для повышения специфичности связывания CRISPR/nCas9 и CRISPR/dCas9 с целевыми последовательностями ДНК. Способ повышения специфичности связывания CRISPR/nCas9 и CRISPR/dCas9 комплексов, согласно настоящему изобретению, имеет важное значение для создания клеточных биомедицинских продуктов и генной терапии наследственных заболеваний человека. 5 з.п. ф-лы, 7 ил., 1 пр.
1. Биомолекулярный комплекс для избирательного связывания участков двухцепочечной молекулы ДНК, включающий два Cas-белка и две молекулы РНК, каждая из которых состоит из трех фрагментов:
1) гидовой РНК (гРНК), способной связываться с Cas-белком и целевой молекулой ДНК,
2) димеризующегося домена, способного связывать две молекулы РНК между собой, и
3) линкерного фрагмента РНК, способного соединять гидовую РНК и димеризующийся домен.
2. Комплекс по п. 1, характеризующийся тем, что для его построения выбирают такой тип взаимного расположения Cas-белков, при котором расстояние между кислородами, расположенными на 3' конце гРНК двух Cas-белков, будет наименьшим, при этом структуры Cas-белков и их гРНК стерически не взаимодействуют, а также отсутствуют стерические препятствия для построения РНК-фрагментов - 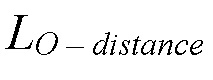 .
.
3. Комплекс по п. 1, характеризующийся тем, что структуру димеризующегося домена РНК выбирают на основании его способности к димеризации со своей же копией.
4. Комплекс по п. 3, характеризующийся тем, что выбирают димеризующийся домен РНК, длина которого 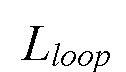 не превышает половины
не превышает половины 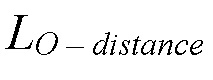 , затем для выбранного домена рассчитывают длину участка, участвующего в димеризации -
, затем для выбранного домена рассчитывают длину участка, участвующего в димеризации - 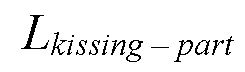 .
.
5. Комплекс по п. 1, характеризующийся тем, что количество нуклеотидов, формирующих линкерный участок, рассчитывают по формуле (1):
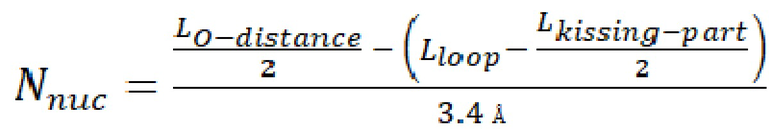 (1),
(1),
где 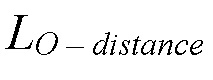 - расстояние между кислородами на 3'-концах гРНК Cas-белков,
- расстояние между кислородами на 3'-концах гРНК Cas-белков, 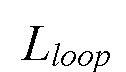 - длина выбранного димеризующегося РНК домена,
- длина выбранного димеризующегося РНК домена, 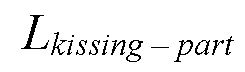 - длина участка, участвующего в kissing-loop взаимодействии, 3,4 Å - константа, описывающая расстояние между парами азотистых оснований в составе РНК.
- длина участка, участвующего в kissing-loop взаимодействии, 3,4 Å - константа, описывающая расстояние между парами азотистых оснований в составе РНК.
6. Комплекс по п. 1, характеризующийся тем, что нуклеотидный состав линкерного участка оптимизируют таким образом, чтобы линкерный участок не участвовал в формировании вторичных структур с димеризующимся РНК-фрагментом и участком гРНК.
| WO 2020243365 A1, 03.12.2020 | |||
| WO 2015035162 A2, 12.03.2015 | |||
| WO 2015021426 A1, 12.02.2015 | |||
| RU 2021105208 A, 02.09.2022 | |||
| WILES M.V | |||
| et al | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Genome, 2015, v | |||
| Прибор для получения стереоскопических впечатлений от двух изображений различного масштаба | 1917 |
|
SU26A1 |
Авторы
Даты
2025-03-06—Публикация
2023-12-30—Подача