Предполагаемое изобретение относится к области медицинской микробиологии и генетической инженерии и может быть использовано в лабораторной диагностике при проведении мониторинга за холерой, планировании противоэпидемических мероприятий и исследований, связанных с изучением особенностей биологии возбудителя.
Неотъемлемой частью идентификации штаммов холерных вибрионов при их выделении от людей либо из объектов окружающей среды является оперативное определение их эпидемической опасности по наличию генов холерного токсина (ctxA) и токсин - корегулируемых пилей адгезии (tcpA), которое проводится с использованием метода ПЦР (МУК 4.2.3745-22). Описано два типа гена tcpA у V.cholerae «классический», обнаруживаемый у вибрионов классического биовара, вызвавшего шестую пандемию холеры, и tcpA типа Эль Тор, характерный для вибрионов Эль Тор - возбудителей текущей седьмой пандемии холеры (1).
В настоящее время для идентификации основных факторов патогенности холерного вибриона с помощью ПЦР в формате реального времени в Российской Федерации предложен ряд способов: тест-система «Амплисенс Vibrio cholerae-FL» (Регистрационное удостоверение: № ФСР 2011/11139), позволяющая определить видовую принадлежность штамма к виду V. cholerae, серогруппе и обнаружить гены ctxA и tcpA типа Эль Тор, а также способ молекулярного типирования типичных (O1) и атипичных (RO) нетоксигенных штаммов V.cholerae El Tor с помощью ПЦР в режиме реального времени (4). Указанные способы обеспечивают возможность выявлять в ПЦР ген tcpA типа Эль Тор и используются в практике работы лабораторий при мониторинге холеры.
Однако в ходе мониторинга холеры в 2023 году от людей выделено два нетоксигенных штамма V.cholerae серовара O1 Эль Тор, которые при проведении ПЦР с зарегистрированной тест-системой «Амплисенс Vibrio cholerae-FL» и по другому методу (4) дали отрицательный результат на наличие гена tcpA. Тем не менее, проведенное полногеномное секвенирование этих культур и последующий анализ с помощью программы для анализа результатов полногеномного секвенирования V.cholerae «SeqAnalyzer» (5) показало наличие в их геномах гена tcpA Classical типа, отличающегося на 81,3% от референс - последовательности гена tcpA Эль Тор. Данное отличие в нуклеотидной последовательности объясняет отрицательный результат при выявления у V.cholerae гена tcpA Classical типа с помощью используемых в настоящее время способов ПЦР-детекции.
Наиболее современным способом выявления генов является полногеномное секвенирование изучаемого штамма V.cholerae с последующим биоинформационным анализом результатов и сравнением с базами данных с помощью пакета программ. Однако данный прием занимает несколько дней, требует работы высококвалифицированного персонала, амортизации дорогостоящих секвенаторов, использования картриджей, расходных материалов, беспрепятственного доступа к международным базам данных. Поэтому данный прием не может широко применяться при оперативном анализе большого числа культур и при необходимости быстрого получения ответа.
Наиболее близким способом выявления гена tcpA является ПЦР в формате реального времени. Однако недостатком указанного способа выявления гена tcpA Classical типа является высокая степень генетического различия, которая не позволяет выявлять ген tcpA Classical типа с помощью наборов праймеров, используемых в данных способах (3,4).
Техническим результатом предлагаемого изобретения является разработка простого, быстрого и достоверного способа выявления гена tcpA Classical типа V.cholerae с помощью ПЦР в формате реального времени.
Поставленная задача достигается тем, что в известном способе выявления гена tcpA Classical типа V.cholerae с помощью ПЦР, включающем выделение ДНК из исследуемого штамма, постановку ПЦР со специфическими праймерами и последующим учетом реакции амплификации в формате реального времени, отличие заключается в том, что амплификацию исследуемой ДНК проводят с парой праймеров:
ген tcpCA 565 - CACAGGCTCAATGCAAGACAC
ген tcpCA 964 - CCATCCCTAATACGCAGAAGC
при этом в инкубационную смесь вносят 0,5 мкл интеркалирующего красителя SYBR Green I для выявления продуктов амплификации по динамике изменения показателя флюоресценции по каналу FAM амплификатора проводят учет результатов по геометрическому методу путем регистрации показателя Ср. При наличии целевого продукта гена tcpA Classical типа V.cholerae формируется целевой ампликон с низким Ср, а при отсутствии специфической мишени, реакция с парой праймеров tcpCA 565 и tcpCA 964 образует амплификат с более высоким показателем Ср (Таблица 2).
При этом ПЦР проводят в объеме 25 мкл и реакционная смесь содержит: 2,5 мкл буфера, 1 Eд Taq ДНК - полимеразы, 0,2 мкМ смеси дНТФ, 0,5 мкл 50Х SYBR Green I, 1,0 мкМ каждого из праймеров tcpCA 565 и tcpCA 964, 5 мкл ДНК матрицы, оставшийся объем - вода.
Кроме того ПЦР проводят с соблюдением режимов: денатурация при 94°С - 2 мин (1 цикл); затем 35 циклов: денатурация при 94°С - 8 секунд, отжиг и учет по каналу FAM при 60°С - 12 секунд.
Обоснование выбора праймеров.
Важнейшим этапом при разработке ПЦР, обеспечивающим получение корректного результата, является правильный подбор мишеней для посадки праймеров. Праймеры для выявления гена tcpA Classical типа Vibrio cholerae были сконструированы авторами с помощью комплекса программного обеспечения, разработанного во ФКУЗ Ростовский-на-Дону противочумный институт Роспотребнадзора г. Ростов-на-Дону (Таблица 1).
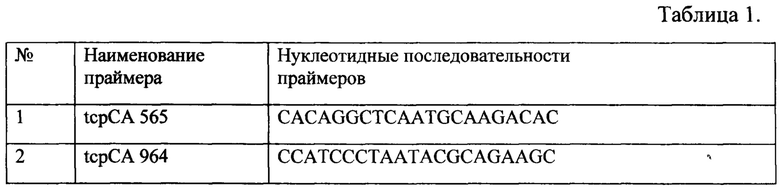
Объектом защиты настоящего изобретения является набор праймеров tcpCA 565 и tcpCA 964, способ проведения реакции и учета результатов для выявления гена tcpA Classical типа Vibrio cholerae помощью ПЦР - РВ с использованием интекалирующего красителя SYBR Green I.
Препарат ДНК из исследуемого штамма анализируют с помощью ПЦР-РВ с набором праймеров tcpCA 565 и tcpCA 964. Использование интекалирующего красителя SYBR Green I необходимо для выявления продуктов амплификации по величине Ср регистрируемой по каналу FAM амплификатора.
Дизайн разработанных праймеров делает ненужным использование внутреннего контроля, используемого для оценки присутствия различных ингибиторов. Решаемая задача достигается тем, что при наличии целевого продукта-гена tcpA Classical типа V.cholerae формируется ампликон с низким Ср в пределах 7-12 цикла, а при отсутствии специфической мишени реакция происходит с образованием других ампликонов с Ср в диапазоне 20-25 (Таблица 2). Если в пробе присутствует ингибитор амплификации, то нарастания флюоресценции не происходит, и показатель Ср не регистрируют.

При этом ПЦР проводят в объеме 25 мкл и реакционная смесь содержит: 2,5 мкл буфера, 1 Ед Taq ДНК - полимеразы, 0,2 мкМ смеси дНТФ, 0,5 мкл 50Х SYBR Green I, 1,0 мкМ каждого из праймеров tcpCA 565 и tcpCA 964, 5 мкл ДНК матрицы, оставшийся объем - вода.
Кроме того ПЦР проводят с соблюдением режимов: денатурация при 94°С - 2 мин (1 цикл); затем 35 циклов: денатурация при 94°С - 8 секунд, отжиг и учет по каналу FAM при 60°С - 12 секунд.
Способ осуществляется следующим образом.
Перед постановкой ПЦР проводят предварительную подготовку материала (выделение ДНК). Культуру, после выращивания из плотной питательной среды в течение 18-24 часов, стерильной палочкой перемещают в пробирку, содержащую 3-5 мл физиологического раствора. Доводят плотность суспензии до 109 микробных клеток на мкМ/мл среды по оптической плотности, после чего выделяют ДНК для постановки ПЦР с помощью любого коммерческого набора для выделения ДНК.
С полученной таким образом ДНК проводят реакцию амплификации с набором праймеров tcpCA 565 и tcpCA 964 в присутствии интеркалирующего красителя SYBR Green I.
Условия проведения реакции в формате реального времени.
Инкубационная смесь объемом 25 мкл содержит: 2,5 мкл буфера, 1 Ед Taq ДНК-полимеразы, 0,2 мкМ смеси дНТФ, 0,5 мкл 50Х SYBR Green I (производства ЗАО Евроген, Москва), 1,0 мкМ каждого праймеров tcpCA 565 и tcpCA 964, 5 мкл ДНК матрицы, оставшийся объем - вода.
Амплификацию и последующее плавление ампликонов проводят в амплификаторе, например, DT-lite5 производства НПФ ДНК-технология, по следующей схеме: денатурация при 94°С - 2 мин (1 цикл); затем 35 циклов: денатурация при 94°С - 8 секунд, отжиг и учет по каналу FAM при 60°С - 12 секунд. Учет реакции проводят по каналу FAM.
Сущность изобретения поясняется следующими примерами.
Все штаммы взяты из лаборатории «Коллекция патогенных микроорганизмов» ФКУЗ Ростовский-на-Дону противочумный институт Роспотребнадзора.
Пример 1. Выявление гена tcpA Classical типа с помощью ПЦР в режиме реального времени у V.cholerae El Tor
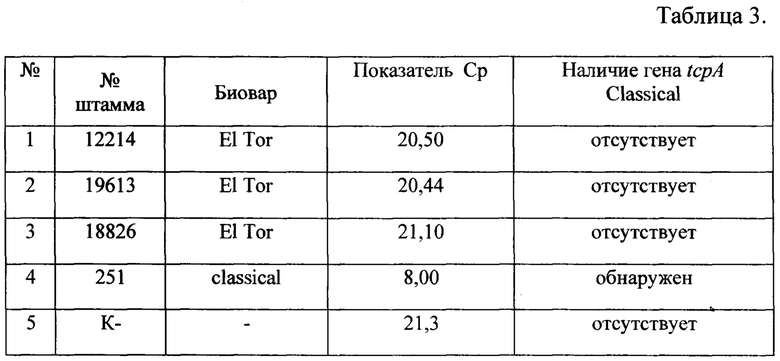
Анализ музейных штаммов V.cholerae O1 El Tor 12214, 19613, 18826 и V.cholerae O1 classical 251 хранящихся в коллекции ФКУЗ Ростовский-на-Дону противочумный институт Роспотребнадзора.
Суточные агаровые культуры суспендировали в дистиллированной воде до 1 × 109 м.кл/мл и выделяли ДНК с помощью набора «РИБОПРЕП». В качестве негативного контроля (К-) использовали дистиллированную воду. Затем проводили постановку ПЦР с учетом результатов в формате реального времени с выделенной ДНК в амплификаторе ДТ-Lite5 (ДНК-технология, Россия) с использованием предложенных нами праймеров. Амплификацию проводили по следующей схеме: денатурация при 94°С - 2 мин (1 цикл); затем 35 циклов: денатурация при 94°С - 8 секунд, отжиг и учет по каналу FAM при 60°С - 12 секунд. Учет реакции проводили по каналу FAM.
Таким образом, из данных Таблицы 3 следует, что ген tcpA Classical типа отсутствует у штаммов V.cholerae O1 El Tor 12214, 19613, 18826, выделенных в ходе седьмой пандемии и обнаружен только у V.cholerae O1 classical 251 - возбудителя шестой пандемии холеры. В случае использования отрицательного контроля (K-) показатель Ср составил 21,3, что свидетельствовало об успешном прохождении ПЦР и отсутствии в пробе ингибиторов амплификации.
Пример 2. Выявление гена tcpA Classical типа с помощью ПЦР в режиме реального времени у V. cholerae биоваров El Tor и classical
Получение препаратов ДНК, приготовление реакционных смесей, проведение помощью ПЦР в режиме реального времени, и учет результатов проводили как в Примере 1, результаты представлены в Таблице 4.
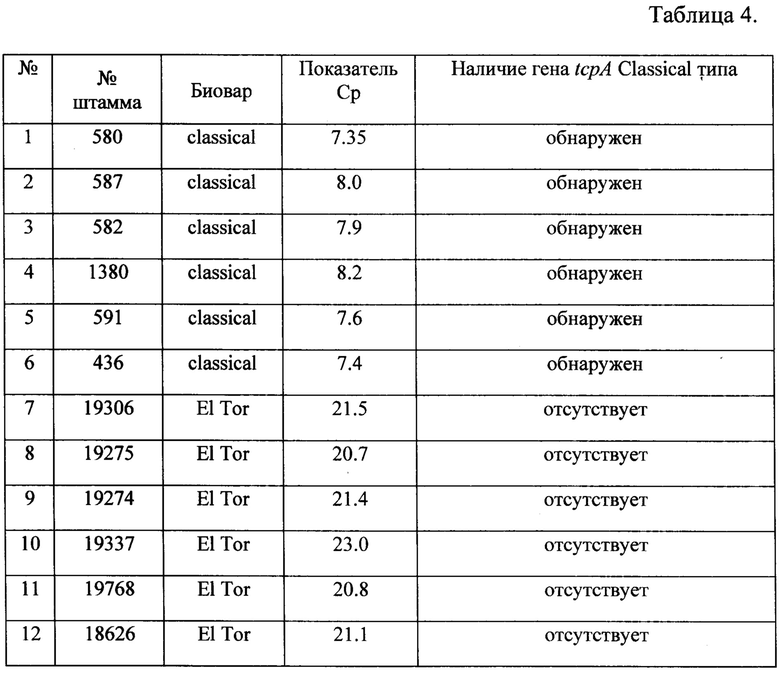
Полученные данные представлены в таблице 4. Штаммы были сгруппированы в соответствии с генетической характеристикой. Из данных Таблицы 4 следует, что ген tcpA Classical типа обнаружен только у музейных штаммов V. cholerae O1 classical, выделенных в ходе шестой пандемии холеры (580, 587, 582, 1380, 591, 436) отсутствует у штаммов V. cholerae O1 El Tor 19306,19275, 19274, 19337, 19768, 18626, выделенных в ходе седьмой пандемии.
Таким образом, из данных Таблиц 3 и 4 следует, что предлагаемый способ позволяет выявить ген tcpA Classical типа у штаммов и V.cholerae O1 El Tor.
Пример 3. Выявление гена tcpA Classical типа с помощью ПЦР в режиме реального времени у двух свежевыделенных штаммов V.cholerae El Tor 226 и 228.
Получение препаратов ДНК, приготовление реакционных смесей, проведение помощью ПЦР и учет результатов проводили как в Примере 1, результаты представлены в Таблице 5.
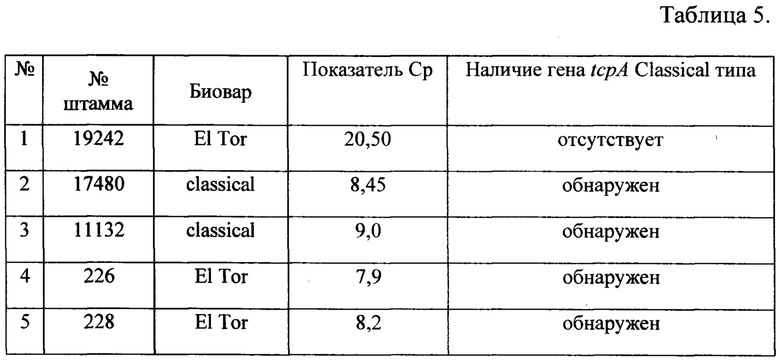
Проведен анализ музейных штаммов V. cholerae O1 El Tor 19242, V.cholerae O1 classical 17480 и 11132, а также двух свежевыделенных в ходе мониторинга холеры в 2023 году штаммов V. cholerae O1 El Tor 226 и 228, хранящихся в коллекции ФКУЗ Ростовский-на-Дону противочумный институт Роспотребнадзора.
При анализе полученных результатов установлено, что предложенный способ детектирует отсутствие гена tcpA Classical типа у музейной культуры V.cholerae O1 El Tor 19242, а также выявляет наличие гена tcpA Classical типа у двух штаммов V.cholerae O1 El Tor 226 и 228, выделенных выходе мониторинга холеры в 2023 году. Полученные результаты свидетельствуют о приобретении штаммами V.cholerae O1 El Tor 226 и 228 седьмой пандемии генетической детерминанты гена tcpA Classical типа, характерной для штаммов шестой пандемии.
Таким образом, наличие гена tcpA Classical типа у двух штаммов V. cholerae O1 El Tor 226 и 228, выделенных в ходе мониторинга холеры в 2023 году, свидетельствует о генетическом обмене детерминанты патогенности между штаммами холерного вибриона шестой и седьмой пандемий. Более того, данный факт невозможно выявить при использовании зарегистрированной тест-системы «Амплисенс Vibrio cholerae-FL» и другими методами (3,4).
Использование предполагаемого изобретения позволяет за счет сконструированных праймеров, проводить выявление гена tcpA Classical типа у V.cholerae O1 El Tor методом полимеразной цепной реакции в режиме реального времени. Это позволит достоверно, быстро и эффективно проводить выявление важной детерминанты патогенности.
Кроме того, простое в исполнении тестирование, невысокая себестоимость, специфичность данных праймеров, отсутствие необходимости в импортных реактивах даст возможность применять их в практике бактериологических лабораторий центров гигиены и эпидемиологии, лечебно-профилактических, противочумных и других учреждений.
Источники информации
1. Kumar A, Das В, Kumar N. Vibrio pathogenicity Island-1: the master determinant of Cholera pathogenesis. Front Cell Infect Microbiol. 2020 Oct 6;10:561296. doi: 10.3389/fcimb.2020.561296.
2. МУК 4.2.3745-22 «Методы лабораторной диагностики холеры.» Утверждены Федеральной службой по надзору в сфере защиты прав потребителей и благополучия человека 12 мая 2022 г.
3. Набор реагентов для выявления ДНК Vibrio cholerae и идентификации патогенных штаммов Vibrio cholerae в биологическом материале и объектах окружающей среды методом полимеразной цепной реакции (ПЦР) с гибридизационно-флуоресцентной детекцией "АмплиСенс® Vibrio cholerae-FL" по ТУ 9398-058-01897593-2012. ФСР 2011/11139 от 13.03.2019
4. Способ молекулярного типирования типичных (O1) и атипичных (RO) нетоксигенных штаммов V. choleraeEYTor с помощью ПЦР в режиме реального времени. Патент RU 2792156 С1. Опубликовано: 17.03.2023 Бюл. №8.
5. SeqAnalyzer-программа для анализа результатов полногеномного секвенирования Vibrio cholerae, определения кратности вариабельных тандемных повторов (VNTR) и выявления INDEL-маркеров. Свидетельство о государственной регистрации программы для ЭВМ №2017613695, РФ; 2016, 24 марта 2017.
--->
<ST26SequenceListing dtdVersion="V1_3" fileName="Способ выявления
гена tcpA Classical V.cholerae El Tor с помощью ПЦР в режиме
реального времени.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2024-02-21">
<ApplicationIdentification>
<IPOfficeCode/>
<ApplicationNumberText/>
<FilingDate>2024-02-21</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>200</ApplicantFileReference>
<ApplicantName languageCode="ru">ФКУЗ Противочумный институт
Роспотребнадзора</ApplicantName>
<ApplicantNameLatin>FKYZ Protivochymni institut
Rospotrebnadzora</ApplicantNameLatin>
<InventorName languageCode="ru">Водопьянов Сергей
Олегович</InventorName>
<InventorNameLatin>Vodopyanov Sergei Olegovich</InventorNameLatin>
<InventionTitle languageCode="ru">
Способ выявления гена tcpA Classical V.cholerae EL Tor с помощью ПЦР
в режиме реального времени
</InventionTitle>
<SequenceTotalQuantity>2</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacaggctcaatgcaagacac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccatccctaatacgcagaagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Данное изобретение относится к области медицинской микробиологии. Описан способ выявления гена tcpA Classical типа у V. cholerae El Tor с помощью ПЦР в режиме реального времени, включающий выделение ДНК из исследуемого штамма, проведение ПЦР с парой разработанных праймеров с последующим учетом реакции амплификации. Технический результат заключается в разработке простого, быстрого и достоверного способа выявления гена tcpA Classical типа V. cholerae с помощью ПЦР в формате реального времени. 2 з.п. ф-лы, 5 табл., 3 пр.
1. Способ выявления гена tcpA Classical типа V. cholerae El Tor с помощью ПЦР в режиме реального времени, включающий выделение ДНК из исследуемого штамма, проведение ПЦР с последующим учетом реакции амплификации, отличающийся тем, что амплификацию исследуемой ДНК проводят с парой праймеров:
tcpCA 565 CACAGGCTCAATGCAAGACAC
tcpCA 964 CCATCCCTAATACGCAGAAGC,
при этом в инкубационную смесь вносят 0,5 мкл интеркалирующего красителя 50Х SYBR Green1 для выявления продуктов амплификации по динамике изменения показателя флюоресценции по каналу FAM амплификатора, учет результатов осуществляют по геометрическому методу путем регистрации показателя Ср, отраженного на экране монитора, а именно: при наличии целевого продукта гена tcpA Classical типа V. cholerae формируется ампликон с низкой величиной Ср в пределах 7-12 цикла, подтверждая присутствие тестируемого гена, в случае отсутствия специфической мишени реакция с парой праймеров образует амплификон с более высоким показателем Ср в пределах 20-25 цикла, подтверждая отсутствие тестируемого гена.
2. Способ по п. 1, отличающийся тем, что ПЦР проводят в объеме 25 мкл и реакционная смесь содержит: 2,5 мкл буфера, 1 Ед Taq ДНК-полимеразы, 0,2 мкМ смеси дНТФ, 0,5 мкл 50Х SYBR Green I, 1,0 мкМ каждого из праймеров, 5 мкл ДНК матрицы, оставшийся объем - вода.
3. Способ по п. 1, отличающийся тем, что ПЦР реакции проводят с соблюдением режимов: денатурация при 94°С - 2 мин, 1 цикл; затем 35 циклов: денатурация при 94°С 8 секунд, отжиг и учет по каналу FAM при 60°С - 12 секунд.
| СПОСОБ ВЫЯВЛЕНИЯ ТОКСИГЕННЫХ ШТАММОВ 01 VIBRIO CHOLERAE "ПОСТГАИТЯНСКОЙ" ЛИНИИ МЕТОДОМ ПЦР В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ | 2021 |
|
RU2766192C1 |
| Способ молекулярного типирования типичных (O1) и атипичных (RO) нетоксигенных штаммов V. cholerae El Tor с помощью ПЦР в режиме реального времени | 2022 |
|
RU2792156C1 |
| Taylor R | |||
| K | |||
| et al | |||
| A Vibrio cholerae classical TcpA amino acid sequence induces protective antibody that binds an area hypothesized to be important for toxin-coregulated pilus structure | |||
| Infection and immunity, 2004, V.72(10), P | |||
| РАМКА ДЛЯ ХРАНЕНИЯ И УЧЕТА КАРТОЧЕК | 1926 |
|
SU6050A1 |
Авторы
Даты
2025-03-25—Публикация
2024-04-01—Подача