Предполагаемое изобретение относится к областям медицинской микробиологии, биотехнологии и молекулярной биологии, а именно к молекулярной диагностике, и может быть использовано для лабораторной диагностики холеры, как в практическом здравоохранении, так и в научно-исследовательских учреждениях и лабораториях.
В настоящие время остается актуальной проблема заболевания холерой: в мире ежегодно регистрируется до 5 млн случаев, из которых 100-120 тыс. заканчиваются смертельно (1). В связи с этим холера на сегодняшний день остается глобальной проблемой мирового здравоохранения, что определяет необходимость совершенствования лабораторной диагностики данной инфекции.
Особый интерес вызывает проблема возникновения и эволюции возбудителя холеры. Недавно предложена оригинальная гипотеза формирования патогенного возбудителя из свободноживущих нетоксигенных холерных вибрионов, обитающих в водной среде Индостанского субконтинента. Процесс был реализован в два основных этапа: первый заключался в приобретении вибрионами в результате горизонтального переноса генов острова патогенности VPI-1 с генами токсин-корегулируемых пилей (tcp). Следующий этап эволюции - получение дополнительно к VPI-1 второго мобильного элемента - нитчатого фага СТХϕ, несущего гены холерного токсина. Эти события привели к появлению нового патогена и гибели миллионов людей не менее, чем за тысячелетний период существования этой болезни (2).
Согласно предложенной гипотезе среди свободноживущих нетоксигенных холерных вибрионов Индостанского субконтинента, явившихся родоначальниками патогенных вибрионов, отсутствовали варианты, адаптированные к низким температурам, что обусловило отсутствие возможности сохранения сформированных эпидемически опасных клонов в регионах с более холодным климатом, включая Россию (2).
На модели других микроорганизмов установлено, что одним из универсальных механизмов адаптации микроорганизмов к низкой температуре является продукция белков холодового шока (3). Недавно у значительного процента штаммов V.cholerae O1 и неО1/неО139, циркулирующих в Российской Федерации, было зарегистрировано наличие гена холодового шока cshl (4,5). Теоретически данное обстоятельство за счет горизонтального переноса генов подобным csh1-вариантам может привести к появлению новых клонов патогенных вибрионов, приспособленных к выживанию в водоемах при низких температурах. На сегодняшний день отсутствует метод, позволяющий выявлять ген холодового шока cshl. Новый высокоспецифичный метод петлевой изометрической амплификации (LAMP) является простым, быстрым, пригодным для масштабного скрининга и является альтернативным многообещающим инструментом для чувствительного и быстрого обнаружения холерных вибрионов.
Известен способ выявления гена холодового шока cshl у штаммов V.cholerae с помощью ПЦР в режиме реального времени, включающий выделение ДНК из исследуемого штамма, постановку ПЦР со специфическими праймерами с последующим плавлением образовавшихся ампликонов. В этом случае учет проводят по двум параметрам: формирование амплификона с низким Ср и температурой плавления 81-82,5°С (6).
Недостатком данного способа является проведение исследования в два этапа: собственно постановка ПЦР и последующая реакция плавления сформированного ампликона, что удлиняет время получения результата.
Современный уровень развития лабораторной диагностики характеризуется разработкой ускоренных способов альтернативных ПЦР. Одним из таких способов является технология петлевой амплификации LAMP (7), которая заключается в использовании четырех или шести олигонуклеотидных праймеров и фермента Bst-полимеразы, что позволяет значительно повысить специфичность реакции. Данный способ позволяет провести то же исследование, что и с помощью реакции ПЦР, но значительно быстрее, специфичнее, не требует дорогого приборного оснащения и квалифицированного персонала.
Технической задачей предлагаемого изобретения является разработка новых высокоспецифичных олигонуклеотидных праймеров для быстрого, выявления гена холодового шока cshl у штаммов V.cholerae O1 и неО1/неО139 в пробах чистых культур, основанного на петлевой изотермической амплификации фрагмента гена-мишени.
Поставленная задача достигается тем, что в известном способе идентификации и определения патогенности штаммов V.cholerae методом LAMP, включающем выделение ДНК из исследуемого материала, проведение петлевой изометрической амплификации и учет результатов, отличием является то, что амплификацию проводят с помощью набора олигонуклеотидных праймеров к гену cshl следующего состава:
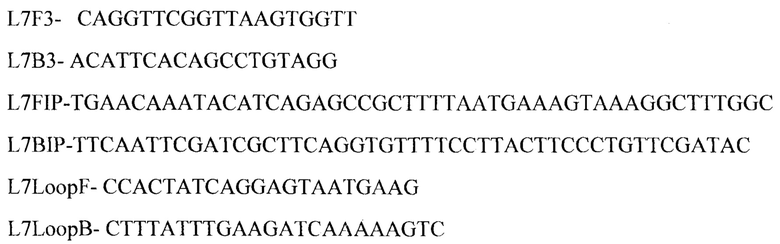
При этом реакцию петлевой изометрической амплификации проводят с помощью амплификатора по схеме двух ступеней: первая 64,5°С, 30 сек; вторая 65°С, 30 сек с учетом по каналу FAM, всего 50 циклов, а детекцию результатов амплификации осуществляют по каналу флуорофора FAM по геометрическому показателю Ср, выводимому в окне результатов амплификатора.
При этом для проведения LAMP предварительно готовят 10-кратную смесь праймеров для каждого из генов, которая включает: 16 мкМ каждого FIP- и BIP-праймеров, 2 мкМ каждого F3- и В3-праймеров, 4 мкМ каждого LoopF- и Loop В-праймеров, вода без нуклеаз до конечного объема 100 мкл, при этом положительный контроль включает в себя все компоненты реакции и препарат ДНК cshl+ штамма V.cholerae, а в случае отрицательного контроля в реакционную смесь вместо исследуемого препарата ДНК вносят 5 мкл деионизованной воды, не содержащей исследуемой ДНК.
Амплификацию проводят в объеме 25 мкл, инкубационная смесь содержит 12,5 мкл буфера БиоМастер LAMP SYBR (2х) (производство Biolabmix, Новосибирск, Россия), 2,5 мкл 10-кратной смеси праймеров, 5 мкл ДНК матрицы, оставшийся объем - вода.
Обоснование выбора праймеров и ДНК-мишени
Объектом защиты настоящего изобретения является набор из шести праймеров L7F3, L7B3, L7FIP, L7BIP, L7LoopF и L7LoopB для выявления гена холодового шока cshl у штаммов V.cholerae O1 и не O1/не O139 с помощью LAMP.
Для конструирования праймеров был использован интернет-сервис Primer Explorer 5 (LAMP primer designing software: http://primerexplorer.jp/e/), BLAST NCBI.
В качестве мишени использовали нуклеотидную последовательность штамма V.cholerae O1 20000 ctxA- tcpA- (GenBank:CP036500.1). Сконструированные праймеры имели следующую структуру:
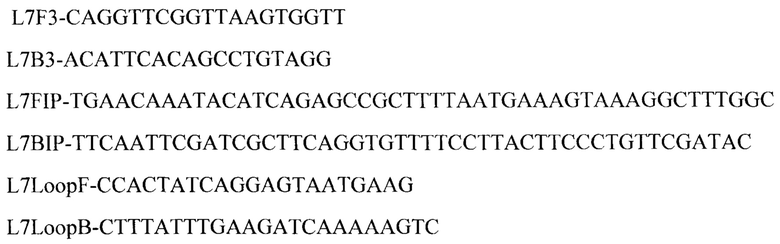
При изучении нуклеотидной последовательности штамма V.cholerae O1 20000 ctxA- tcpA- (NCBI accession number CP036500.1) в составе второй хромосомы идентифицирован ген холодового шока, обозначенный заявителем как cshl.
Cshl является членом семейства белка CspA, локализован в позиции 846905-846693, его размер составляет 213 нуклеотидов. Ген cshl присутствует в штаммах V. cholerae и широко используется в качестве регулирования синтеза различных клеточных белков, как при пониженных значениях температуры, так и в оптимальных для роста температурных условиях.
Способ осуществляется следующим образом:
Этапы:
1. Приготовление смеси праймеров L7F3, L7B3, L7FIP, L7BIP, L7LoopF и L7LoopB.
2. Подготовка исследуемых культур, включая контрольные штаммы и выделение ДНК.
3. Подготовка и проведение реакции петлевой изотермической амплификации (LAMP).
4. Учет результатов.
Первоначально готовят 10-кратную смесь праймеров, которая включает: 16 мкМ каждого FIP- и BIP-праймеров, 2 мкМ каждого F3- и В3-праймеров, 4 мкМ каждого LoopF- и Loop В-праймеров, вода без нуклеаз до конечного объема 100 мкл. Полученную смесь хранят при -20°С в течение 12 месяцев.
Работу проводят в соответствии с требованиями МУ 1.3.2569-09 и СанПиН 3.3686-21 (8,9). Перед постановкой ПЦР проводят предварительную подготовку материала (выделение ДНК). Бактериальную массу, сформировавшую газон на плотной питательной среде, стерильной палочкой перемещают в пробирку, содержащую 3-5 мл физиологического раствора. Доводят плотность суспензии до 109 клеток на мл среды по оптической плотности по стандарту мутности бактериальных взвесей ОСО 42-28-86-2018 (ФГБУ «НЦЭСМП» Минздрава России) после чего выделяют ДНК для постановки ПЦР с помощью любого коммерческого набора для выделения ДНК. С полученной таким образом ДНК проводят реакцию амплификации с праймерами L7F3, L7B3, L7FIP, L7BIP, L7LoopF и L7LoopB.
Условия проведения реакции LAMP для выявления гена холодового шока cshl у штаммов V.cholerae O1 и неО1/неО139: инкубационная смесь объемом 25 мкл содержит 12,5 мкл буфера БиоМастер LAMP SYBR (2х) (производство Biolabmix, Новосибирск, Россия), 2,5 мкл 10-кратной смеси праймеров, 5 мкл ДНК матрицы, оставшийся объем - вода. В качестве негативного (К-) контроля используют деионизованную воду.
Амплификацию проводят в амплификаторе, например, DTlite5 производства НПФ ДНК-технология, по схеме двух ступеней первая: 64,5°С, 30 сек; вторая: 65°С, 30 сек с учетом по каналу FAM, всего 50 циклов. Таким образом, реакция занимает 50 минут.
Сущность изобретения поясняется примерами
Пример 1
Все штаммы взяты из коллекции патогенных микроорганизмов ФКУЗ Ростовский-на-Дону противочумный институт Роспотребнадзора.
Определение чувствительности реакции LAMP для выявления гена холодового шока cshl.
Бактериальную массу cshl+клеток V.cholerae 20000, сформировавших газон на плотной питательной среде, стерильной палочкой перемещают в пробирку, содержащую 3-5 мл физиологического раствора. Доводят плотность суспензии до 109 клеток на мл среды по оптической плотности по стандарту мутности бактериальных взвесей ОСО 42-28-86-2018 (ФГБУ «НЦЭСМП» Минздрава России). Из полученной суспензии готовят десяткратные разведения на физиологическом растворе и выделяют ДНК для постановки ПНР. В качестве негативного контроля (К-) используют деионизованную воду.
Инкубационная смесь объемом 25 мкл содержит: 12,5 мкл буфера БиоМастер LAMP SYBR (2х) (производство Biolabmix, Новосибирск, Россия), 2,5 мкл 10-кратной смеси праймеров, 5 мкл ДНК матрицы, оставшийся объем - вода.
Амплификацию проводят в амплификаторе, например, DTlite5 производства НПФ ДНК-технология, по схеме двух ступеней - первая: 64,5°С, 30 сек; вторая: 65°С, 30 сек с учетом по каналу FAM, всего 50 циклов.
Вывод: результат, оцениваемый по геометрическому показателю Ср, выводимому в окне результатов амплификатора (Таблица 1) показывает, что положительный результат регистрируется в пробах №1-5. В пробах №6-8 результат отрицателен. В пробе №9 (отрицательный контроль) результат отрицателен. Положительный результат в пробе №5 (содержит 104 cshl+ клеток V.cholerae) и отрицательный в пробах №6-8 (содержат большие разведения целевой матрицы), свидетельствует, что чувствительность реакции LAMP для выявления гена холодового шока cshl составляет 104 бактериальных клеток в мл. Время получения результата составило менее 30 минут.
Пример 2
Выявление гена холодового шока cshl с помощью реакции LAMP у штаммов V.cholerae 01, выделенных в ходе мониторинга водных объектов в 2023 году в Ростовской области.
В ходе мониторинга за холерой в Ростовской области в 2023 году выделено пять штаммов V.cholerae O1. Наличие антигена O1 подтвердили проведением ПЦР с набором реагентов «АмплиСенс V.cholerae-FL». С помощью LAMP проводим выявление гена холодового шока cshl. В качестве положительного контроля (К+) использовали cshl+ клетки V.cholerae 20000. Условия культивирования, подготовки проб и проведения реакции LAMP как в Примере 1.
Результаты исследования пяти штаммов V.cholerae O1, выделенных в ходе мониторинга в 2023 году в Ростовской области, представлены в Таблице 2. Положительный результат, оцениваемый по показателю Ср, получен с образцами ДНК из штаммов 65, 66, 84 и образцом положительного контроля. В случае штаммов 63 и 83 и отрицательного контроля результат отрицателен. Полученные результаты свидетельствуют, что штаммы 65, 66 и 84 содержат ген холодового шока cshl. Время получения результата составило менее 30 минут.
Пример 3
Выявление гена холодового шока cshl с помощью реакции LAMP у штаммов V.cholerae не O1/неО139, выделенных в ходе мониторинга в 2023 году в Ростовской области.
В ходе мониторинга за холерой в Ростовской области в 2023 году выделено девять штаммов V.cholerae не O1/неО139. Отсутствие антигена O1 подтвердили проведением ПЦР с набором реагентов «АмплиСенс V.cholerae-FL».
Результаты исследования девяти штаммов V.cholerae неО1/неО139, выделенных в ходе мониторинга в 2023 году в Ростовской области, представлены в Таблице 3. Положительный результат, оцениваемый по показателю Ср, получен с образцами ДНК из штаммов 28, 35, 38, 40(41-2), 41 (41-3) и с образцом положительного контроля (К+). В случае исследования штаммов 34, 39, 44, 49 и отрицательного контроля результат отрицателен. Полученные данные свидетельствуют, штаммы 28, 35, 38, 40 (41-2) и 41 (41-3) содержат ген холодового шока cshl. Время получения результата составило менее 30 минут.
Использование предлагаемого изобретения позволяет за счет набора шести высокоспецифичных праймеров L7F3, L7B3, L7FIP, L7BIP, L7LoopF и L7LoopB быстро в течение не более 30 минут, проводить в реакции LAMP выявление гена холодового шока cshl у штаммов V.cholerae O1 и не O1/не O139.
Кроме того, простота в исполнении анализа, низкая себестоимость, высокая чувствительность (1x104 м. кл. /мл) дает возможность применять его в практике бактериологических лабораторий центров гигиены и эпидемиологии, лечебно-профилактических, противочумных и других учреждений.
Источники информации
1. Всемирная организация здравоохранения (16 декабря 2022 г.). Новости о вспышках болезней. Холера - ситуация в мире. См. по адресу: https://www.who.int/ru/emergencies/disease-outbreak-news/item/2022-DON426
2. Сергиев В.П., Кутырев В.В. Холера и гибель древнейшей Индской цивилизации. Проблемы особо опасных инфекций. 2023; 2:95-100. DOI: 10.21055/0370-1069-2023-2-95-100
3. Barria С., Malecki М., Arraiano С.М. Bacterial adaptation to cold. Microbiology. 2013;159():2437-2443. https://doi.org/10.1099/mic.0.052209-0
4. Бородина O.B., Водопьянов C.O., Водопьянов A.C., Олейников И.П., Чемисова O.C, Полеева M.B. Изучение встречаемости гена холодового шока cshl у штаммов Vibrio cholerae, циркулирующих на территории Российской Федерации // Бактериология. - 2021. - Т. 6. - №3. - С. 22-23.
5. Водопьянов C.O., Бородина О.В., Ежова М.И., Олейников И.П., Водопьянов А.С., Носков А.К. Ген холодового шока cshl у Vibrio cholerae nonO1/nonO139. Вестник биотехнологии и физико-химической биологии им. Ю.А. Овчинникова 2022; 18(4):12-19.
6. Водопьянов C.O., Бородина О.В., Водопьянов А.С., Олейников И.П. Патент «Способ выявления гена холодового шока cshl у штаммов Vibrio cholerae с помощью ПЦР в режиме реального времени». Патент на изобретение №2783023 от 08.11.2022.
7. Notomi Т., Okayama Н., Masubuchi Н. et al. Loop-mediated isothermal amplification of DNA // Nucleic Acids Res. - 2000. - Vol. 28. - e63-e63. DOI: 10.1093/nar/28.12.e63.
8. СанПиН 3.3686-21. Санитарно-эпидемиологические требования по профилактике инфекционных болезней.
9. Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности: методические указания МУ 1.3.2569-09.-М.; 2009.

--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3" fileName="не
О139 с помощью методом петлевой изотермической амплификации
(LAMP).xml" softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2023-08-01">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>191</ApplicationNumberText>
<FilingDate>2023-08-01</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>191</ApplicantFileReference>
<ApplicantName languageCode="ru">Ростовский- на –Дону противочумный
институт Роспотребнадзора</ApplicantName>
<ApplicantNameLatin>Rostov-on-Don Plague Control Research Institute
of the Rospotrebnadzor</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ выявления гена холодового
шока csh1 у штаммов Vibrio cholerae О1 и неО1/не О139 с помощью
методом петлевой изотермической амплификации (LAMP).</InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caggttcggttaagtggtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acattcacagcctgtagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value> vibrio cholerae </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgaacaaatacatcagagccgcttttaatgaaagtaaaggctttggc</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcaattcgatcgcttcaggtgttttccttacttccctgttcgatac</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccactatcaggagtaatgaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctttatttgaagatcaaaaagtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ оценки внутривидовой конкуренции токсигенных и нетоксигенных штаммов Vibrio cholerae О1 и О139 серогрупп с помощью ПЦР в режиме реального времени | 2023 |
|
RU2806564C1 |
| СПОСОБ ВЫЯВЛЕНИЯ ГЕНА ХОЛОДОВОГО ШОКА CSH1 У ШТАММОВ VIBRIO CHOLERAE С ПОМОЩЬЮ ПЦР В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ | 2021 |
|
RU2783023C1 |
| Способ выявления гена tcpA Classical V. cholerae El Tor с помощью ПЦР в режиме реального времени | 2024 |
|
RU2837089C1 |
| Способ идентификации и определения патогенности штаммов Vibrio cholerae методом петлевой изотермической амплификации (LAMP) | 2023 |
|
RU2820141C1 |
| Способ молекулярного типирования типичных (O1) и атипичных (RO) нетоксигенных штаммов V. cholerae El Tor с помощью ПЦР в режиме реального времени | 2022 |
|
RU2792156C1 |
| Способ выявления токсигенных штаммов О1 Vibrio cholerae "гаитянской" группы методом ПЦР в режиме реального времени по конечной точке | 2019 |
|
RU2729218C1 |
| СПОСОБ ВЫЯВЛЕНИЯ ТОКСИГЕННЫХ ШТАММОВ 01 VIBRIO CHOLERAE "ПОСТГАИТЯНСКОЙ" ЛИНИИ МЕТОДОМ ПЦР В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ | 2021 |
|
RU2766192C1 |
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОГО ВНУТРИВИДОВОГО ТИПИРОВАНИЯ V. cholerae О1 И О139 СЕРОГРУПП | 2014 |
|
RU2575046C2 |
| Способ обнаружения жизнеспособных холерных вибрионов 01 серогруппы биоваров Classical и El Tor в окружающей среде с помощью бактериофага M3 методом количественной ПЦР | 2023 |
|
RU2808577C1 |
| Способ молекулярно-генетического внутривидового типирования токсигенных штаммов Vibrio cholerae О1 Eltor | 2016 |
|
RU2627193C1 |
Изобретение относится к областям медицинской микробиологии, биотехнологии и молекулярной биологии. Сущность изобретения заключается в том, что амплификацию проводят с помощью набора из шести праймеров к гену cshl следующего состава:
Реакцию петлевой изометрической амплификации проводят с помощью амплификатора по схеме двух ступеней - первая: 64,5°С, 30 сек; вторая: 65°С, 30 сек с учетом по каналу FAM, всего 50 циклов, а детекцию результатов амплификации осуществляют по каналу флуорофора FAM по геометрическому показателю Ср, выводимому в окне результатов амплификатора. Для проведения LAMP предварительно готовят 10-кратную смесь праймеров для каждого из генов, которая включает: 16 мкМ каждого FIP- и BIP- праймеров, 2 мкМ каждого F3- и В3-праймеров, 4 мкМ каждого LoopF- и Loop В-праймеров, вода без нуклеаз до конечного объема 100 мкл. Положительный контроль включает в себя все компоненты реакции и препарат ДНК cshl+ штамма V.cholerae. Отрицательный контроль - деионизованная вода, не содержащая исследуемой ДНК. Амплификацию проводят в объеме 25 мкл и инкубационная смесь содержит: 12,5 мкл буфера БиоМастер LAMP SYBR (2х) (производство Biolabmix, Новосибирск, Россия), 2,5 мкл 10-кратной смеси праймеров, 5 мкл ДНК матрицы, оставшийся объем - вода. Изобретение расширяет арсенал средств молекулярной диагностики и может быть использовано для лабораторной диагностики холеры как в практическом здравоохранении, так и в научно-исследовательских учреждениях и лабораториях. 2 з.п. ф-лы, 3 табл., 3 пр.
1. Способ выявления гена холодового шока cshl у штаммов V.cholerae О1 и неО1/неО139 методом петлевой изотермической амплификации (LAMP), включающий выделение ДНК из исследуемого материала, проведение петлевой изометрической амплификации и учет результатов, отличающийся тем, что амплификацию проводят с помощью набора из шести праймеров к гену cshl следующего состава:
L7F3 CAGGTTCGGTTAAGTGGTT
L7B3 ACATTCACAGCCTGTAGG
L7FIP TGAACAAATACATCAGAGCCGCTTTTAATGAAAGTAAAGGCTTTGGC
L7BIP TTCAATTCGATCGCTTCAGGTGTTTTCCTTACTTCCCTGTTCGATAC
L7LoopFCCACTATCAGGAGTAATGAAG
L7LoopBCTTTATTTGAAGATCAAAAAGTC
при этом реакцию петлевой изометрической амплификации проводят с помощью амплификатора по схеме двух ступеней - первая: 64,5°С, 30 сек; вторая: 65°С, 30 сек с учетом по каналу FAM, всего 50 циклов, а детекцию результатов амплификации осуществляют по каналу флуорофора FAM по геометрическому показателю Ср, выводимому в окне результатов амплификатора.
2. Способ по п. 1, отличающийся тем, что для проведения LAMP предварительно готовят 10-кратную смесь праймеров для каждого из генов, которая включает: 16 мкМ каждого FIP- и BIP-праймеров, 2 мкМ каждого F3- и В3-праймеров, 4 мкМ каждого LoopF- и Loop В-праймеров, вода без нуклеаз до конечного объема 100 мкл, при этом положительный контроль включает в себя все компоненты реакции и препарат ДНК cshl+ V.cholerae, а в случае отрицательного контроля в реакционную смесь вместо исследуемого препарата ДНК вносят 5 мкл деионизованной воды, не содержащей исследуемой ДНК.
3. Способ по п. 1, отличающийся тем, что амплификацию проводят в объеме 25 мкл и инкубационная смесь содержит: 12,5 мкл буфера БиоМастер LAMP SYBR 2х, 2,5 мкл 10-кратной смеси праймеров, 5 мкл ДНК матрицы, оставшийся объем - вода.
| Способ идентификации нетоксигенных штаммов холерных вибрионов О1 серогруппы с помощью ПЦР для выделения генетических детерминант | 2017 |
|
RU2665542C1 |
| Способ молекулярного типирования типичных (O1) и атипичных (RO) нетоксигенных штаммов V. cholerae El Tor с помощью ПЦР в режиме реального времени | 2022 |
|
RU2792156C1 |
| WO 2022095921 A1, 12.05.2022 | |||
| Е.В | |||
| Монахова, И.В | |||
| Архангельская | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Токарный резец | 1924 |
|
SU2016A1 |
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
Авторы
Даты
2024-05-14—Публикация
2023-08-29—Подача