Изобретение относится к области биотехнологии, а именно медицинской микробиологии и может быть использовано в лабораторной диагностике для обнаружения жизнеспособных вибрионов Vibrio cholerae. в пробах окружающей среды путем применения бактериофа М3.
В настоящее время существует проблема отдельных вспышек холеры не только в эндемичных регионах, но и в других местах при завозных случаях из-за свободной миграции населения. Часто возникают эпидемии в странах, пострадавших от массовых стихийных бедствий (1).
Бактериофаги используются в практике для дифференциации бактерий из-за высокой специфичности, что обусловило их прицельное инфицирующее действие даже на отдельные штаммы в пределах вида бактерий. В результате этого, возможность получения ложноположительного результата обнаружения холерных вибрионов минимальна (2, 3).
На сегодняшний день «золотым стандартом» анализа жизнеспособности бактерий считается культуральный метод, который показывает наблюдаемое деление отдельной клетки на колонии на чашках с агаром или в жидкой среде, тем самым доказывая, что клетки живы (4).
Количественная полимеразная цепная реакция (количественная ПЦР) - это средство измерения концентрации ДНК или количества копий определенного гена или генетической области в образце по отношению к известному набору стандартных концентраций ДНК или калибраторов в случае относительного количественного определения, и это достигается путем оценки в реальном времени количества ДНК, реплицируемого во время реакции ПЦР.
Для ускорения выдачи ответа и ухода от культуральных процедур подтверждения жизнеспособности бактерий при ДНК-анализе предложен подход с использованием для этой цели формата ПЦР в реальном времени (ПЦР-РВ) с гибридизационно флуоресцентной детекцией, позволяющего проводить количественную оценку ДНК мишени в процессе амплификации. Выбор такого подхода был обоснован возможностью установить разницу в уровнях накопления ампликонов при исследовании обогащаемых проб, содержащих живые и неживые бактерии.
Известна тест-система Ампли Сенс® Vibrio cholerae-FL (5), позволяющая выявить холерные вибрионы (Vibrio cholerae) в различных образцах, но по ее результатам нельзя судить о жизнеспособности микроба. В результате при исследовании параллельно используют культуральные методы, получая чистую культуру, для физического доказательства присутствия живых холерных вибрионов, что увеличивает время диагностики и возможности быстрого принятии решения по методу лечения.
За прототип выбран способ определения жизнеспособности и количественный анализ патогенов, переносимых водой, путем обогащения (6), заключающийся в том, что проводят обогащение концентрата бактерий из пробы с последующим взятием первого экстракта ДНК в момент времени Т0 и второго экстракта ДНК в момент времени Т2 после инкубационного периода, при этом ПЦР выполняют на эктрактах ДНК получая соответствующие значения порога цикла Ct, а изменение Кт указывает на целевой патоген в образце.
Однако несмотря на применение количественного ПЦР, с помощью данного способа невозможно распознавать живые холерные вибрионы в пробах окружающей среды.
Технической задачей предполагаемого изобретения является быстрое и достоверное обнаружение жизнеспособных холерных вибрионов в пробах окружающей среды с помощью бактериофага М3 на основе количественной ПЦР, что позволит уйти от длительных культуральных методов исследования и приведет к ускорению выдачи результата.
Поставленная задача достигается тем, что в способе обнаружения жизнеспособных холерных вибрионов 01 серогруппы биоваров Classical и El Tor в окружающей среде с помощью бактериофага М3 методом количественной ПЦР, полимеразную цепную реакцию проводят в реальном времени с детекцией концентрации ДНК бактериофага М3 лизирующего холерные вибрионы 01 серогруппы биоваров Classical и El Tor, с использованием праймеров и зонда:
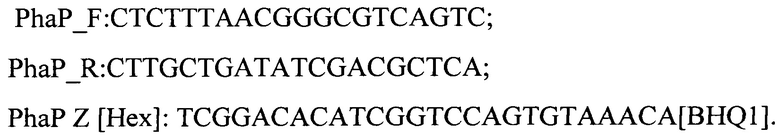
при этом учет результатов ПЦР проводят по геометрическому методу Ср путем регистрации на стандартной калибровочной кривой в течение заданных промежутков времени, более 2-х часов, если наблюдают накопление вирусных частиц бактериофага М3, то обнаруживают присутствие в пробах жизнеспособных холерных вибрионов 01 серогруппы биоваров Classical и El Tor, а в случае отсутствия накопления вирусных частиц подтверждают их нежизнеспособность.
При этом для выделения ДНК бактериофага М3 предварительно перед ПЦР культивируют в 10,0 мл пептонного бульона четырех часовую пробу в объеме 0,2 мл, затем отбирают 100 мкл в эппендорф с добавлением 0,3 мл фага М3 в титре 102 БОЕ/мл, перемешивают, отбирают 100 мкл в эппендорф (1,5 мл), что соответствует нулевой точке (Т0), смесь размещают в термостат при 37°С на один час, затем два часа, а после выделяют стандарты ДНК от 108 до 103 БОЕ/мл комплектом реагентов.
Кроме того, ПЦР проводят в объеме 25 мкл и реакционная смесь содержит: 2,5 мкл буфера, 1 Eд Taq ДНК-полимеразы, 0,2 мкМ смеси дНТФ,), 1,0 мкМ каждого из праймеров PhaP_F, PhaP_R и, 0,5 мкМ зонда PhaPZ, 5 мкл ДНК-ДНК матрицы, оставшийся объем - вода.
При этом ПЦР проводят с соблюдением следующих этапов и режимов:
денатурация при 94°С - 2 мин (1 цикл); затем 35 циклов: денатурация при 94°С - 8 секунд, отжиг и учет по каналу HEX при 60°С - 12 секунд.
В качестве материала для исследования выбран холерный бактериофаг М3, который был выделен из внешней среды (воды). Фаг хранится в коллекции-депозитарии лаборатории бактериофагов ФКУЗ Ростовский-на-Дону противочумный институт Роспотребнадзора.
Бактериофаг М3 зарегистрирован и доступен в международной базе Genbank (NCBI): MN379460.1-MN379463.1.
Технический результат достигается благодаря биологическим свойствам бактериофага, а именно: литической активности - 83,3%, скорости адсорбции, латентный период - 47 мин., урожайности (Burstsize), и генетической характеристики нуклеотидных последовательностей. Для бактериофага М3 были сконструированы зонд и специфические праймеры, с помощью которых осуществляют детектирование увеличение матрицы ДНК. Проводят подготовку стандартов фаговой ДНК от 108 до 103 БОЕ/мл (БОЕ означает «бляшко образующая единица») для калибровочной кривой при учете результатов в ПЦР.
Специфичность в отношении клетки-хозяина подтверждена на большом наборе представителей близкородственных микроорганизмов семейств Vibrionaceae, Pseudomonadaceae, Enterobacteriaceae. Анализ нуклеотидных последовательностей показал, что размер генома бактериофага М3 составляет 46669 п.н., включающий 50 ORF, и все они принадлежат роду Vibrio. Генетические детерминанты факторов антибиотико резистентности, токсинов и интеграз не обнаружены, что подтверждает вирулентную природу фага.
Обоснование выбора праймеров.
Важнейшим этапом при разработке ПЦР, обеспечивающим получение корректного результата, является правильный подбор мишений для посадки праймеров. С помощью комплекса программного обеспечения https://bioinfo.ut.ee/primer3-0.4.0/, разработанного в ФКУЗ РостНИПЧИ Роспотребнадзора (г.Ростов-на-Дону), были созданы праймеры на фаг М3 для проведения ПЦР. Для этого в программу вносили последовательность бактериофага и зонда, который был сконструирован вручную, далее получены прямой и обратный праймеры:
Сконструированная система из праймеров (PhaP_F, PhaP_R) и зонда (PhaP Z) была специфична для бактериофага М3, поскольку в ПЦР с другими образцами фагов (PhageYersiniaEnterocolytica, Yersiniapseudotuberculosis, Vibriometschnikovii, Vibriomimicus, Vibrioparahaemolyticus, Vibrio cholerae) были получены отрицательные результаты.
Способ осуществляется следующим образом.
Перед постановкой ПЦР проводят подготовку материала (выделение ДНК).
Для этого осуществлялют по методике (Габрилович, 1968), размножение фага. В 10 мл пептонного бульона добавлялют 0,2 мл 4-часовой пробы из окружающей среды (индикаторная культура). Отбирают 100 мкл в эппендорф (объемом 1,5 мл), что служит отрицательным контролем.
Затем проводят культивирование пробы, для этого добавляют 0,3 мл фага М3 в титре 102 БОЕ/мл, перемешивают и сразу отбирают 100 мкл в следующий эппендорф 1,5 мл. Данную пробу считают нулевой точкой (Т0). Смесь помещают в термостат при температуре 37°С, и проводят лизис через 1 час культивирования с отбором 100 мкл в эппендорфы 1,5 мл (T1). Затем лизис через 2 часа культивирования также с отбором 100 мкл в эппендорфы 1,5 мл (Т2) и так далее.
Выделяют ДНК с помощью комплекта реагентов «РИБО-преп» АмплиСенс® согласно инструкции производителя.
Таким образом готовя стандарты фаговой ДНК (от 108 до 103 БОЕ/мл) детектируют увеличение матрицы ДНК.
С полученной ДНК далее проводят реакцию амплицикации с набором праймеров и зондом.
Условия проведения реакции ПЦР-РВ.
Инкубационная смесь объемом 25 мкл содержит: 2,5 мкл буфера, 1 Ед Taq ДНК-полимеразы, 0,2 мкМ смеси дНТФ, (производства ЗАО Евроген, Москва), 1,0 мкМ каждого из праймеров PhaP_Р, PhaP_R и, 0,5 мкМ зонда PhaPZ, 5 мкл ДНК-ДНК матрицы, оставшийся объем - вода.
Амплификацию проводят в амплификаторе, например, (DTlite5 производства НПФ ДНК-технология), по следующей схеме: денатурация при 94°С - 2 мин (1 цикл); затем 35 циклов: денатурация при 94°С - 8 секунд, отжиг и учет по каналу HEX при 60°С - 12 секунд.
Учет результатов проводят геометрическим методом путем регистрации на показателей Ср, используя в качестве стандартов разведения фага М3, содержащие 104, 105, 106 БОЕ/мл. Если происходит нарастание количества вирусных частиц указанного бактериофага М3 после инкубирования пробы в течение нескольких временных приемов не менее двух часов, то делают заключение о присутствии в пробе жизнеспособных холерных вибрионов O1 серогруппы биоваров Classical и ElTor.
Сущность предполагаемого изобретения поясняется примерами.
Пример 1.
Обнаружение накопленных частиц фага М3 после размножения на живой культуре штамма V.cholerae 145 взятого из коллекции музея Ростовского-на-Дону противочумного института Роспотребнадзора.
Осуществляют способ по вышеуказанной технологии. Титрование проводят в пептоном бульоне. Отбирают по 100 мкл каждого разведения в эппендорфы (1,5 мл). Выделение ДНК осуществляют комплектом реагентов «РИБО-преп» АмплиСенс®.
Подготовленные стандарты фаговой ДНК (от 108 до 103 БОЕ/мл) используют в качестве стандартов при детектировании накопления целевой ДНК. Результат ПЦР выражают в числе фаговых частиц на мл образца (БОЕ/мл) и величиной Ср (см. таблицу 1).
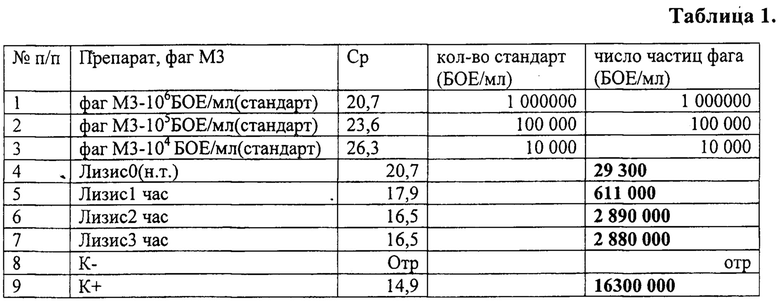
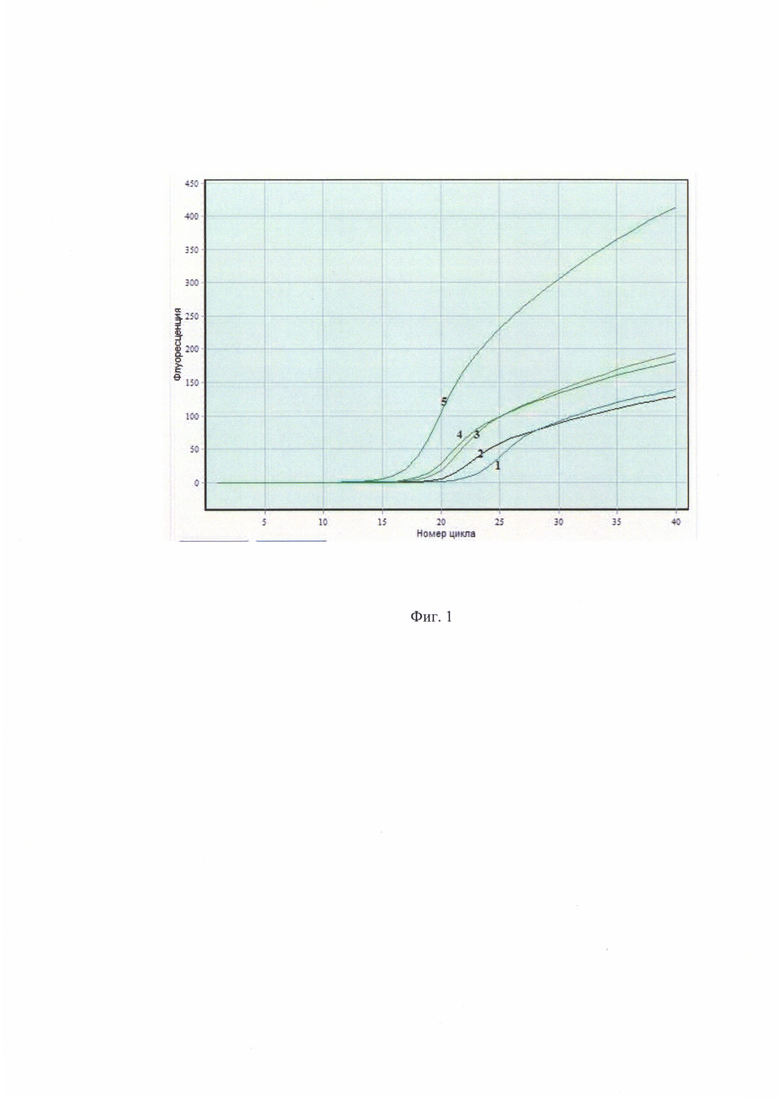
Из таблицы 1 видно, что в качестве стандартов для калибровочной кривой использовали разведения фага М3 - 106, 105 104 БОЕ/мл (строки 1-3). Ср увеличивается на 3 единицы или в 10 раз БОЕ/мл. Остальные разведения фага не информативны. Идет нарастание количества частиц фага, оцениваемое по снижению величины Ср, после 1 (лизис 1 час) и 2 часов (лизис 2 часа) инкубирования в пептоне на индикаторной культуре V.cholerae, а после 3 часов (лизис 3 часа) показатель выходит на плато (строка 7).
Фиг. 1 иллюстрирует нарастание количества частиц фага, оцениваемое по снижению величины Ср. На графике: 1 - лизис 0 (нулевая точка), 2 - лизис через 1 часа инкубации, 3 - лизис через 2 часа, 4 - лизис через 3 часа, 5 - контрольный образец фага. Видно, что, начиная с первого часа инкубации (кривая 2), имеет место нарастание количества фаговых частиц, что проявляется в более раннем подъеме кривой по сравнению с нулевой точкой (кривая 1).
Вывод
Учет результатов проводят геометрическим методом путем регистрации на калибровочной кривой показателей Ср, используя в качестве стандартов разведения фага М3, содержащие 104, 105, 106 БОЕ/мл. Если происходит нарастание количества вирусных частиц указанного бактериофага М3 после инкубирования пробы в течение нескольких временных приемов не менее двух часов, то делают заключение о присутствии в пробе жизнеспособных холерных вибрионов 01 серогруппы биоваров Classical и ElTor (см. фиг. 1).
Пример 2. Применение заявленного способа на прогретых культурах (нежизнеспособных) штаммов V.cholerae биоваров Classical и ElTor отобранных из коллекции музея Ростовского-на-Дону противочумного института Роспотребнадзора: №№145, 438, 1029) и ElTor (штаммы: 20554, 20570, 20556).
Технология проведения способа такая, как в примере 1.
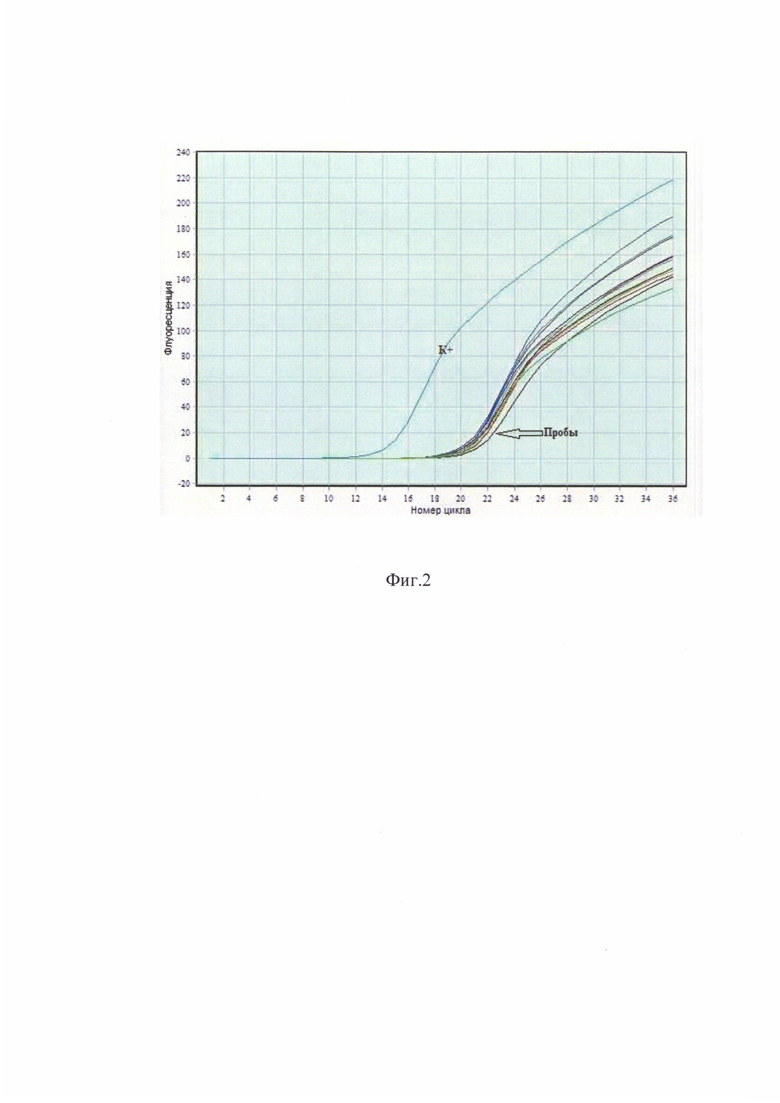
При помощи тест-системы АмплиСенс® Vibrio cholerae-FL в ПЦР-РВ были обнаружены мертвые клетки холерного вибриона, полученные путем прогревания образцов при 65°С в течение часа. Для дополнительного контроля прогретые культуры были проверены на жизнеспособность путем посева на чашки Петри с питательным агаром. Рост V.cholerae не выявлен.
При постановке ПЦР заявленным способом с мертвыми клетками V.cholerae не обнаружено нарастание частиц фага после 2 часов инкубирования при 37°С относительно нулевой точки. Результат ПЦР с убитыми культурами V.Cholerae Eltor(1-3) и VCholerae classical (4-6) см.таблицу 2.
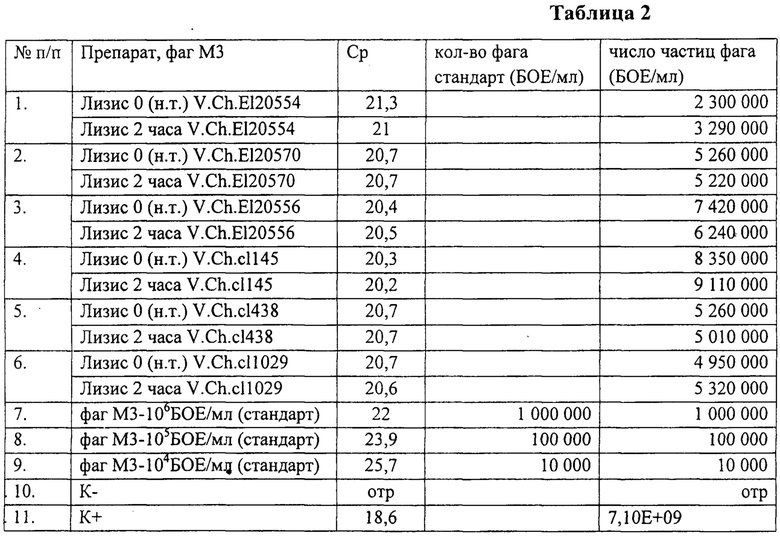
Из таблицы видно, что Ср не увеличивается при лизисе после двух часов инкубирования у всех 6 нежизнеспособных культур (строки 1-6). В качестве стандартов использовали разведения фага 104, 105, 106 БОЕ/мл (строки 7-9).
Вывод
При наличии в пробе нежизнеспособных клеток Vibrio cholerae не регистрируют накопление специфической ДНК фага М3 после инкубирования пробы в течение нескольких временных приемов не менее двух часов. Делается заключение об отсутствии в пробе жизнеспособных холерных вибрионов 01 серогруппы биоваров Classical и ElTor (см. фиг. 2).
Использование предполагаемого изобретения позволяет быстро, в течение трех часов, достоверно определять присутствие в пробе взятой из окружающей среды, жизнеспособных холерных вибрионов 01 серогруппы биоваров Classical и El Tor. Это достигается при помощи бактериофага М3 на основе количественной ПЦР в реальном времени.
Кроме того предложенный способ дает возможность уйти от культуральных методов исследований холеры в клинических лабораториях (до 3-х суток), что значительно сократит время постановки диагноза инфекционного заболевания и назначить курс лечения
Источники информации
1. Centers for Disease Control and Prevention. 2010. Update: cholera outbreak - Haiti, 2010. MMWRMorb. Mortal. Wkly. Rep.59:1473-1479.
2. Коровкина Г.И. и др. Исследование экспериментальных фагов для идентификации холерных вибрионов биовараэльтор // Состояние и перспективы разработки медицинских средств защиты от поражающих факторов радиационной, химической и биологической природы. - 2019. - С. 173-174.
3. Гаевская Н.Е. и др. Способ идентификации холерных вибрионов О1 серогруппы биоваров Classical и El Tor.
4. Bogosian G, Bourneuf EV. A matter of bacterial life and death. EMBO Rep.2001;2(9):770-4.
5. тест-система Ампли Сенс® Vibrio cholerae-FL(рег.уд. №ФСР 2011/11139) https://www.amplisens.ru/upload/iblock/384/FSR%202011 11139 13.03.19. pdf
6. «Способ определения жизнеспособности и количественный анализ патогенов, переносимых водой, путем обогащения», пат.№US 20200208200 A1, кл. C12Q 1/689, 02.07.2020 г.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="en"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Способ обнаружения жизнеспособных холерных вибрионов О1
серогруппы биоваров Classical и El Tor в окружающей среде с помощью
бактериофага М3 методом количественной ПЦР..xml" softwareName="WIPO
Sequence" softwareVersion="2.2.0" productionDate="2023-02-10">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>184</ApplicationNumberText>
<FilingDate>2023-02-10</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>184</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>184</ApplicationNumberText>
<FilingDate>2023-02-10</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Ростовский- на –Дону противочумный
институт Роспотребнадзора</ApplicantName>
<ApplicantNameLatin>Rostov-on-Don Plague Control Research Institute
of the Rospotrebnadzor</ApplicantNameLatin>
<InventorName languageCode="ru">Погожова Марина Павловна
</InventorName>
<InventorNameLatin>Pogojova Marina Pavlovna</InventorNameLatin>
<InventionTitle languageCode="ru">Способ обнаружения жизнеспособных
холерных вибрионов О1 серогруппы биоваров Classical и El Tor в
окружающей среде с помощью бактериофага М3 методом количественной
ПЦР.</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctctttaacgggcgtcagtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttgctgatatcgacgctca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Vibrio cholerae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcggacacatcggtccagtgtaaaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления гена tcpA Classical V. cholerae El Tor с помощью ПЦР в режиме реального времени | 2024 |
|
RU2837089C1 |
| Способ идентификации холерных вибрионов O1 серогруппы биоваров Classical и El Tor | 2019 |
|
RU2729575C1 |
| Способ профилактики холеры с помощью бактериофагов | 2021 |
|
RU2783000C1 |
| Способ идентификации штаммов VIBRIO CHLERAE O1, определения их токсигенности и биовара с дифференциацией биовара ЭльТор на типичные и генетически измененные варианты методом мультиплексной полимеразной цепной реакции и тест-система для его осуществления с учетом результатов в режиме "реального времени" | 2019 |
|
RU2732448C1 |
| Способ идентификации Vibrio cholerae 01 серогруппы биовара El Tor | 2022 |
|
RU2797369C1 |
| СПОСОБ ИДЕНТИФИКАЦИИ ТОКСИГЕННЫХ ШТАММОВ V. CHOLERAE O1, ОПРЕДЕЛЕНИЯ ИХ БИОВАРА И ДИФФЕРЕНЦИАЦИИ ШТАММОВ БИОВАРА ЭЛЬТОР НА ТИПИЧНЫЕ И ИЗМЕНЕННЫЕ МЕТОДОМ МУЛЬТИПЛЕКСНОЙ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ И ТЕСТ-СИСТЕМА ДЛЯ ЕГО ОСУЩЕСТВЛЕНИЯ | 2011 |
|
RU2458141C1 |
| Рекомбинантная плазмида, экспрессирующая клонированный ген шаперона HFQ Vibrio cholerae, и штамм Escherichia coli - суперпродуцент шаперона HFQ Vibrio cholerae | 2018 |
|
RU2707525C1 |
| СПОСОБ ОДНОВРЕМЕННОЙ ИДЕНТИФИКАЦИИ ТОКСИГЕННЫХ ШТАММОВ ГЕНОВАРИАНТОВ ВОЗБУДИТЕЛЯ ХОЛЕРЫ ЭЛЬ ТОР И ИХ ДИФФЕРЕНЦИАЦИИ ПО ЭПИДЕМИЧЕСКОМУ ПОТЕНЦИАЛУ МЕТОДОМ МУЛЬТИПЛЕКСНОЙ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2014 |
|
RU2560280C2 |
| Способ молекулярного типирования типичных (O1) и атипичных (RO) нетоксигенных штаммов V. cholerae El Tor с помощью ПЦР в режиме реального времени | 2022 |
|
RU2792156C1 |
| Способ идентификации токсигенных генетических вариантов возбудителя холеры Эль Тор с набором мутаций в генах вирулентости и генах острова пандемичности VSPII методом мультиплексной полимеразной цепной реакции | 2022 |
|
RU2815711C2 |
Изобретение относится к биотехнологии, медицинской микробиологии и может быть использовано в лабораторной диагностике для обнаружения жизнеспособных вибрионов Vibrio cholerae в пробах окружающей среды. Предложен способ обнаружения жизнеспособных холерных вибрионов 01 серогруппы биоваров Classical и El Tor в окружающей среде. Проводят полимеразную цепную реакцию в реальном времени с детекцией концентрации ДНК бактериофага М3 лизирующего холерные вибрионы 01 серогруппы биоваров Classica и El Tor, с использованием праймеров и зонда. Учет результатов ПЦР проводят по геометрическому методу Ср путем регистрации на стандартной калибровочной кривой в течение заданных промежутков времени, более 2-х часов, если наблюдают накопление вирусных частиц бактериофага М3, то обнаруживают присутствие в пробах жизнеспособных холерных вибрионов 01 серогруппы биоваров Classical и El Tor, а в случае отсутствия накопления вирусных частиц подтверждают их нежизнеспособность. 0Для выделения ДНК бактериофага М3 предварительно перед ПЦР культивируют в 10,0 мл пептонного бульона четырех часовую пробу в объеме 0,2 мл, затем отбирают 100 мкл в эппендорф с добавлением 0,3 мл фага М3 в титре 102 БОЕ/мл, перемешивают, отбирают 100 мкл в эппендорф (1,5 мл), что соответствует нулевой точке (Т0), смесь размещают в термостат при 37°С на один час, затем два часа, а после выделяют стандарты ДНК от 108 до 103 БОЕ/мл комплектом реагентов. Способ позволяет осуществить быстрое и достоверное обнаружение жизнеспособных холерных вибрионов в пробах окружающей среды с помощью бактериофага М3 на основе количественной ПЦР, что позволит уйти от длительных культуральных методов исследования и приведет к ускорению выдачи результата. 3 з.п. ф-лы, 2 ил., 2 табл. 2 пр.
1. Способ обнаружения жизнеспособных холерных вибрионов 01 серогруппы биоваров Classical и El Tor в окружающей среде с помощью бактериофага М3 методом количественной ПЦР, характеризующийся тем, что полимеразную цепную реакцию проводят в реальном времени с детекцией концентрации ДНК бактериофага М3 лизирующего холерные вибрионы 01 серогруппы биоваров Classical и El Tor, с использованием праймеров и зонда:
PhaP_F: CTCTTTAACGGGCGTCAGTC;
PhaP_R: CTTGCTGATATCGACGCTCA;
PhaP Z [Hex]: TCGGACACATCGGTCCAGTGTAAACA[BHQ1],
при этом учет результатов ПЦР проводят по геометрическому методу Ср путем регистрации на стандартной калибровочной кривой в течение заданных промежутков времени, более 2-х часов, если наблюдают накопление вирусных частиц бактериофага М3, то обнаруживают присутствие в пробах жизнеспособных холерных вибрионов 01 серогруппы биоваров Classical и El Tor, а в случае отсутствия накопления вирусных частиц подтверждают их нежизнеспособность.
2. Способ по п. 1, отличающийся тем, что для выделения ДНК бактериофага М3 предварительно перед ПЦР культивируют в 10,0 мл пептонного бульона четырехчасовую пробу в объеме 0,2 мл, затем отбирают 100 мкл в эппендорф с добавлением 0,3 мл фага М3 в титре 102 БОЕ/мл, перемешивают, отбирают 100 мкл в эпиндорф (1,5), что соответствует нулевой точке (Т0), смесь размещают в термостат при 37°С на один час, затем два часа, а после выделяют стандарты ДНК от 108 до 103 БОЕ/мл комплектом реагентов.
3. Способ по п. 1, отличающийся тем, что ПЦР проводят в объеме 25 мкл и реакционная смесь содержит: 2,5 мкл буфера, 1 Ед Тaq ДНК-полимеразы, 0,2 мкМ смеси дНТФ, 1,0 мкМ каждого из праймеров PhaP_F, PhaP_R и 0,5 мкМ зонда PhaPZ, 5 мкл ДНК-ДНК матрицы, оставшийся объем - вода.
4. Способ по п. 1, отличающийся тем, что ПЦР проводят с соблюдением следующих этапов и режимов:
денатурация при 94°С - 2 мин (1 цикл); затем 35 циклов: денатурация при 94°С - 8 секунд, отжиг и учет по каналу HEX при 60°С - 12 секунд.
Авторы
Даты
2023-11-29—Публикация
2023-03-22—Подача