Изобретение относится к области биохимии и медицины, в частности к способам измерения или испытания, использующим микроорганизмы; составы или индикаторную бумагу для них; способам получения подобных составов; контролю за условиями в микробиологических или ферментативных процессах.
Современная наука находит подтверждение тому, что все больше патологий ассоциировано с микроорганизмами. Пока это остается областью научного интереса, мы уже разрабатываем тест-систему, которая перенесет этот концепт в область ин-витро диагностики.
В норме у здорового человека кровь стерильна. Вместе с тем имеется несколько исключений: транзиторная бактериями довольно часто на короткое время возникает после экстракции зуба или других зубных манипуляций, а также хирургические вмешательств на слизистых оболочках, контаминированных микроорганизмами, бронхоскопии или катетеризации уретры. Этот тип транзиторной бактериемии обусловлен в основном комменсалами и всегда купируется спонтанно в результате фагоцитоза бактерий в печени или селезенке. Достаточно большое число локальных инфекций часто сопровождается транзиторной бактериемией: открытые раны, язвы, холециститы, менингиты, пневмонии, пиелонефриты. Бактериемии являются характерной чертой некоторых инфекционных заболеваний, например бруцеллез, лептоспироз, брюшной тиф.
Исследования Koren с соавторами в 2011 году, Rafferty с сотрудниками в 2011 году, а также нашими исследованиями было установлено присутствие в атеросклеротических бляшках Propionibacterium acnes. Так как атеросклеротическая бляшка находится в постоянном контакте с кровью, то часть микроорганизмов составляющих микробное сообщество атеросклеротической бляшки, смывается током крови. Нашими исследованиями было установлено присутствие бактерий Propionibacterium acnes в крови пациентов с атеросклерозом. Наш проект нацелен на установление корреляции между присутствием микроорганизмов как в крови, так и в патологически измененной стенке сосуда, атеросклеротической бляшке.
Известен набор реагентов для выявления и идентификации ДНК возбудителя сибирской язвы методом полимеразной цепной реакции в реальном времени "ом-скрин-сибирская язва-рв", представляющий собой расфасованную в стрипованные микропробирки лиофильно высушенную реакционную смесь, состоящую из эквимолярной смеси дезоксинуклеозидтрифосфатов, Taq-ДНК-полимеразы, синтетической ДНК внутреннего положительного контроля, трегалозы, видоспецифических праймеров для индикации хромосомного маркера Bacillus anthracis - фрагмента гена ssp SEQ ID NO 1 (5'-agcaaacgcacaatcagaag-3'), SEQ ID NO 2 (5'-acgtctgtttcagttgcaaattctgtacc-3') и флуоресцентного зонда SEQ ID NO 3 ((R6G)-ctggtgctagcattcaaagcacaaatgct-(BH2)); праймеров для индикации участка гена сарА плазмиды рХO2 SEQ ID NO 4 (5'-tttcaccagcacccacatagtca-3'), SEQ ID NO 5 (5'-cagtaaaagaagccggatttaca-3') и флуоресцентного зонда SEQ ID NO 6 ((ROX)-tggcgaataaccatatga(C-LNA)ggattatg-(BHQ2)); праймеров для индикации участка гена lef плазмиды pXOl SEQ ID NO 7 (5'-tttcaccagcacccacatagtca-3'), SEQ ID NO 8 (5'-cagtaaaagaagccggatttaca-3') и флуоресцентного зонда SEQ ID NO 9 ((FAM)-cgggcggtcatggtgatgtaggtatg-(RTQ1)); праймеров SEQ ID NO 10 (5'-agaactcgtagcgctagctgtag-3'), SEQ ID NO 11 (5'-cgtagaactagctgtagcgct-3') и флуоресцентного зонда SEQ ID NO 12 ((Cy5)-agcggctcctacttctgcagggg-(BHQ2)) внутреннего положительного контроля, а также в отдельных микропробирках: разбавителя; положительного контрольного образца, представляющего лиофильно высушенную смесь из четырех рекомбинантных плазмид pGem-T, содержащих встроенные специфические последовательности ДНК: фрагмент гена ssp, фрагмент гена lef, кодирующего летальный фактор сибиреязвенного микроба (плазмида pXO1), фрагмент гена капсулообразования сарА (плазмида рХO2), специфический фрагмент контрольной ДНК; отрицательного контрольного образца; внутреннего положительного контроля выделения (см. патент №2542395, МПК C12Q 1/68, опубл. 20.02.2015 г.).
Недостатком данного технического решения является относительная дороговизна синтеза флюоресцентного зонда, который по сути является третьим праймером, и вследствие этого повышенная трудоемкость процедуры смешивания смеси.
Известен набор синтетических олигонуклеотидов для выявления днк возбудителя человеческого моноцитарного эрлихиоза Ehrlichia spp. методом полимеразной цепной реакции "в реальном времени", включающий в себя праймеры: 5'-GGGGAAAGA ТТТАТСGCTATTAG-3', 5'-CGGCATAGCTGGАТСAGGCT-3' и пробу: (BHQ1)-5'-CCCACTGCTGCC(FdT) CCCGTAGGAGTCTGG-3'P, где BHQ1 означает присоединенный к 5'-концевому нуклеотиду темновой гаситель флуоресценции, a FdT - флуоресцентный краситель FAM, присоединенный к нуклеотиду Т (см. патент №2415947, МПК C12Q 1/68, C07H 21/00, опубл. 10.04.2011 г.).
Недостатком данного технического решения является относительная дороговизна синтеза пробы, содержащего темновой гаситель и флюоресцентный краситель, и вследствие этого повышенная трудоемкость процедуры смешивания смеси.
Наиболее близкой к заявляемому изобретению по совокупности существенных признаков является способ обнаружения нуклеиновых кислот в реальном времени методом полимеразной цепной реакции и устройство для его осуществления, включающий введение жидких проб, содержащих нуклеиновую кислоту, в реакционные зоны на верхней поверхности теплопроводящей подложки микрочипа; изолирование вводимых проб от атмосферы; взаимодействие нуклеиновой кислоты пробы с компонентами полимеразно-цепной реакции при термоциклировании проб с отводом тепла через внешнюю поверхность микрочипа; флуоресцентное определение изменения количества продуктов полимеразно-цепной реакции в процессе термоциклирования; определение количества исходной нуклеиновой кислоты в пробах по динамике роста флуоресцентного сигнала (см. патент на изобретение №2385940, МПК C12P 19/00, C12Q 1/68, опубл. 10.04.2010 г.).
Недостатком данного изобретения является необходимость специального дорогостоящего оборудования для работы с микрочипами.
Техническим результатом заявленного изобретения является создание простого в применении и эффективного способа количественного определения ДНК бактерий Propionibacterium acnes методом полимеразной цепной реакции в реальном времени, которая позволит исследовать микробную обсемененность атеросклеротических бляшек не бактериологическим методом, а на основе ПЦР, с гибридизационно-флюорересценной детекцией.
Сущностью заявленного технического решения является способ количественного определения ДНК бактерии Propionibacterium acnes методом ПЦР в реальном времени, заключающийся в выделении, очищении тотальной бактериальной ДНК, выполнении полимеразной цепной реакции в режиме реального времени, где в качестве матрицы для ПЦР используют тотальную бактериальную РНК, включающий праймеры:
P.acnes-Pac-guaA
F:5'-TGCTGCCATTCTTTCTCA-3'
R:5'-TACAGTTTCATCCCGATG-3'
Отрицательный контроль - P.acid - HAMP
F: 5' - TTGAGGTGTCCGATGATG-3'
R: 5' - GGAATGGGAGAAGCAGTT-3',
состав смеси ПЦР:
1. Дезоксинуклеотидтрифосфаты 2,5 мМ;
2. 10-кратный ПЦР Буфер;
3. MgCl2, 25 мМ;
4. Taq ДНК - полимераза с ингибирующими активность фермента антителами 5 Е/мкл;
5. Вода обработанная DEPC;
6. ЕvaGreen краситель (50x раствор в воде).
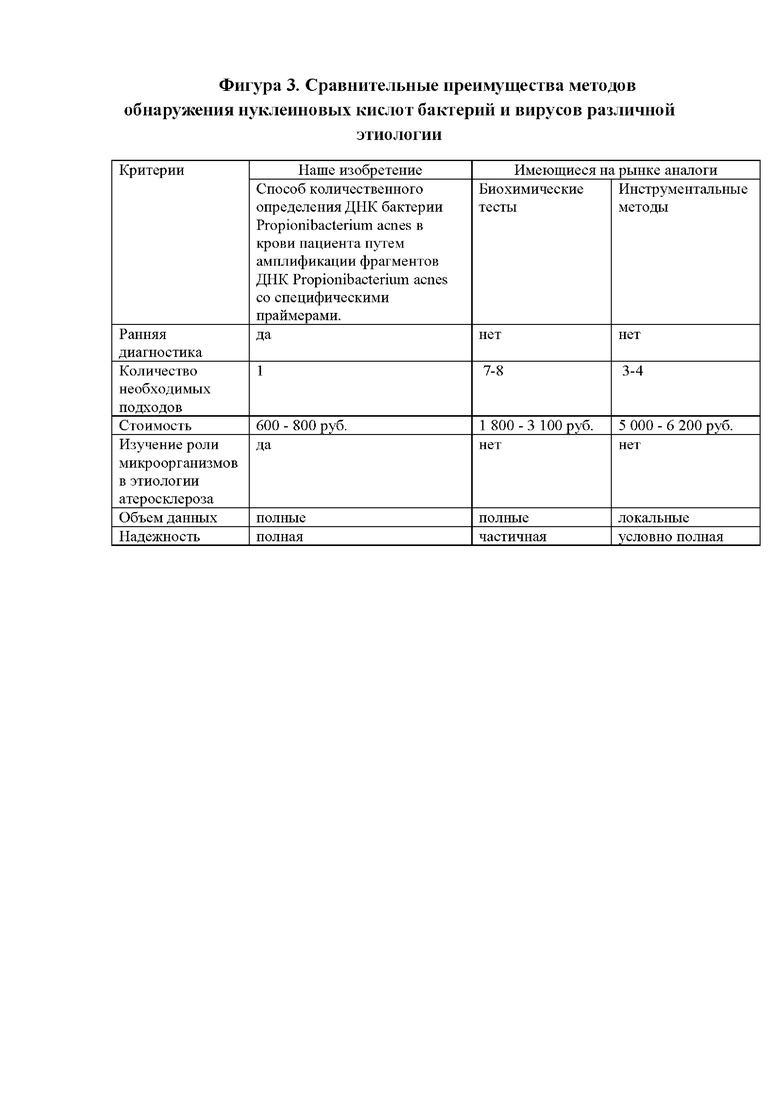
В отличие от аналогичных продуктов, представленных на рынке, результаты заявленного способа не зависят от привередливости микроорганизмов. Метод может быть выполнен в течение одного рабочего дня.
Кроме того, при анализе образцов крови метод позволяет проводить раннюю диагностику атеросклероза сосудов в различных возрастных группах населения и избежать хирургического иссечения атеросклеротических бляшек.
Способ позволит контролировать эффективность терапии, позволит найти корреляцию между качественно-количественным составом микробиомы атеросклератических бляшек и результатами лабораторно-инструментальных методов диагностики атеросклеротических изменений в сосудах. Возможность адаптации метода исследования на другие системы органов человека.
Кроме того, преимуществом предлагаемого изобретения является меньшая стоимость необходимых реагентов, оборудования и трудозатрат персонала по сравнению с конкурирующими решениями.
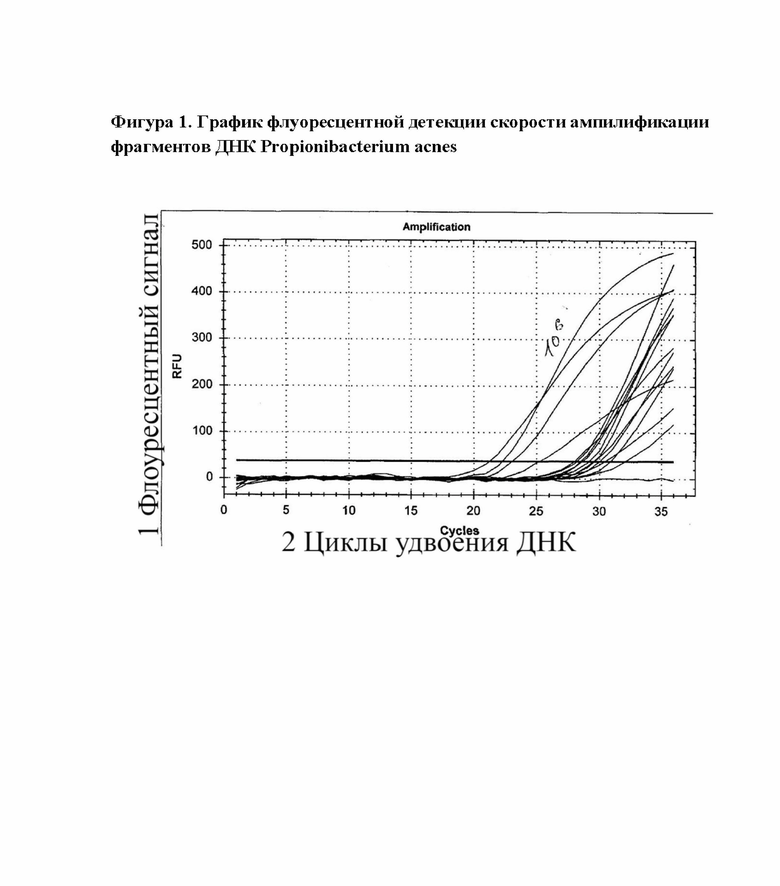
Тем не менее нами были проведены успешные эксперименты по детекции ДНК P.acnes в сыворотках больных (см. фигура 1).
Анализ известных технических решений, проведенный по научно-технической и патентной документации, показал, что совокупность существенных признаков заявляемого изобретения не известна из уровня техники, следовательно, оно соответствует условиям патентоспособности «новизна» и «изобретательский уровень».
Заявляемое изобретение поясняется графическими материалами:
Фигура 1 - График флуоресцентной детекции скорости ампилификации фрагментов ДНК Propionibacterium acnes.
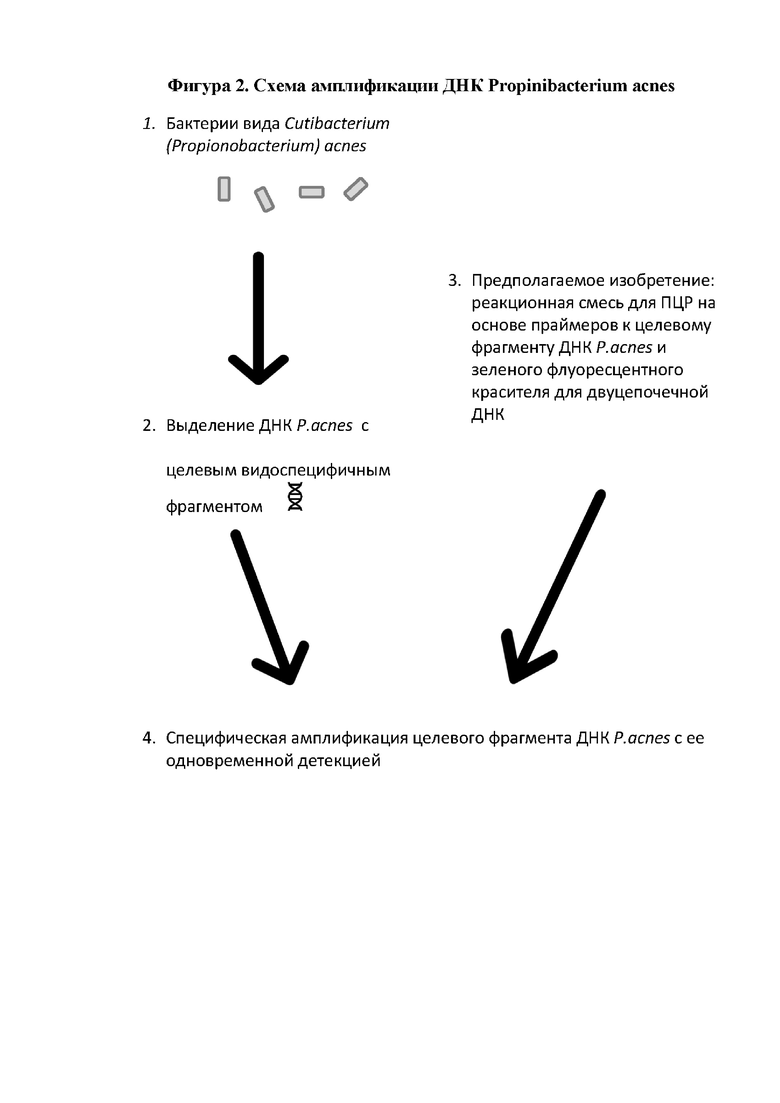
Фигура 2 - Схема амплификации ДНК Propinibacterium acnes.
Фигура 3 - Сравнительные преимущества методов обнаружения нуклеиновых кислот бактерий и вирусов различной этиологии.
Позиции на фигурах обозначают:
Фигура 1: 1 - Флоуресцентный сигнал; 2 - циклы удвоения ДНК.
Фигура 2: 1 - Cutibacterium acnes, 2 - целевой фрагмент ДНК Propinibacterium acnes.
Пара специфично подобранных к ДНК Propionibacterium acnes праймеров используется для амплификации соответствующих праймерам участков ДНК Propionibacterium acnes методом полимеразной цепной реакции (ПЦР). При взаимодействии с двуцепочечной ДНК специальный флюоресцентный краситель, который добавляется с реакционную смесь, при облучении лазером, который находится на борту ПЦР амплификатора, начинает испускать зеленое свечение, которое в свою очередь измеряется специальным светочувствительным детектором ПЦР амплификатора. По мере амплификации ДНК в процессе полимеразной цепной реакции количество двуцепочечных фрагментов увеличивается и вместе с этим увеличивается свечение красителя, что позволяет проводить детекцию амплификации специфичных фрагментов в режиме реального времени, а при наличии соответствующих контролей позволяет определять количество копий ДНК Propionibacterium acnes в изначальном образце.
Принцип работы основан на флуоресцентной детекции скорости ампилификации фрагментов ДНК Propionibacterium acnes при проведении ПЦР.
Далее заявителем приведен пример осуществления заявленного технического решения.
Способ осуществляется следующим образом: проводится параллельная амплифкация фрагментов из ДНК Propionibacterium acnes в исследуемом образце, в положительных контролях с известной концентрацией копий ДНК Propionibacterium acnes, а также в отрицательном контроле, содержащем ДНК бактерий другого вида. На основе анализа амплификационных кривых проводится оценка количества копий ДНК Propionibacterium acnes в исследуемом образце, а также специфичность проведенной реакции.
Заявляемое изобретение соответствует требованию промышленной применимости и может быть осуществлено с применением современных материалов и технологий.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" productionDate="2025-02-18"
originalFreeTextLanguageCode="ru">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2024112319</ApplicationNumberText>
<FilingDate>2024-05-06</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>AB123</ApplicantFileReference>
<ApplicantName languageCode="ru">ГАУЗ Межрегиональный клинико
диагностический центр</ApplicantName>
<ApplicantNameLatin>GAUZ Mezhregionalnyi kliniko diagnosticheskii
tsentr</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ количественного
определения ДНК бактерий Propionibacterium acnes в крови пациента
путем амплификации фрагментов ДНК Propionibacterium acnes со
специфическими праймерами</InventionTitle>
<InventionTitle languageCode="en">Method for quantitative
determination of Propionibacterium acnes DNA in patient's blood by
amplification of Propionibacterium acnes DNA fragments with specific
primers</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Propionibacterium
acnes</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgctgccattctttctca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Propionibacterium
acnes</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacagtttcatcccgatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Propionibacterium
acnes</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgaggtgtccgatgatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Propionibacterium
acnes</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggaatgggagaagcagtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Наборы олигонуклеотидов для выявления ДНК бактерии Helicobacter pylori в клиническом материале методом полимеразной цепной реакции в режиме реального времени | 2024 |
|
RU2839157C1 |
| Тест-система и способ обнаружения специфических фрагментов нуклеиновых кислот 16 патогенов с использованием изотермической реакции амплификации | 2023 |
|
RU2810751C1 |
| Способ выявления возбудителей респираторных инфекций крупного рогатого скота: BPIV, BRSV, BHV-4, BCoV, BVDV-1, BVDV-2, BVDV-3, на основе мультиплексной полимеразной цепной реакции (ПЦР) | 2022 |
|
RU2798286C1 |
| Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых зондов для индикации цереулида и диарейных энтеротоксинов Bacillus cereus complex с помощью ПЦР-РВ | 2024 |
|
RU2839472C1 |
| Способ выявления ДНК микроорганизмов класса Mollicutes в крови и других биоматериалах методом ПЦР-РВ | 2023 |
|
RU2831277C1 |
| Способ индикации цереулида и диарейных энтеротоксинов Bacillus cereus complex c помощью ПЦР-РВ | 2024 |
|
RU2837898C1 |
| Применение набора олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых зондов для индикации цереулида и диарейных энтеротоксинов Bacillus cereus complex с помощью ПЦР-РВ | 2024 |
|
RU2839150C1 |
| Способ идентификации SNP-генотипов возбудителя чумы средневекового биовара филогенетических ветвей 2.MED1 и 2.MED4 из очагов Северного и Северо-Западного Прикаспия методом секвенирования по Сэнгеру | 2024 |
|
RU2831366C1 |
| СПОСОБ ОБНАРУЖЕНИЯ ГЕНОВ УСТОЙЧИВОСТИ К АМИНОГЛИКОЗИДАМ ИЗ ГРУППЫ aadA У БАКТЕРИЙ ЖИВОТНОГО ПРОИСХОЖДЕНИЯ МЕТОДОМ ПЦР С ДЕТЕКЦИЕЙ В РЕЖИМЕ «РЕАЛЬНОГО ВРЕМЕНИ» | 2023 |
|
RU2816522C1 |
| Способ генотипирования однонуклеотидного варианта rs2304277 (G>A) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени | 2024 |
|
RU2836047C1 |
Изобретение относится к области биотехнологии, в частности к способу обнаружения нуклеиновых кислот Propionibacterium acnes в образце крови. Указанный способ осуществляют методом полимеразной цепной реакции в реальном времени с использованием праймеров с последовательностями SEQ ID NO: 1-4, и он включает выделение, очищение тотальной бактериальной ДНК и выполнение полимеразной цепной реакции (ПЦР) в режиме реального времени, а в качестве матрицы для ПЦР используют тотальную бактериальную РНК. Настоящее изобретение обеспечивает эффективный способ определения ДНК бактерий Propionibacterium acnes. 3 ил.
Способ обнаружения нуклеиновых кислот Propionibacterium acnes в образце крови методом полимеразной цепной реакции в реальном времени, включающий выделение, очищение тотальной бактериальной ДНК, выполнение полимеразной цепной реакции (ПЦР) в режиме реального времени, отличающийся тем, что в качестве матрицы для ПЦР используют тотальную бактериальную РНК, предусматривающий использование праймеров:
P.acnes-Pac-guaA
F: tgctgccattctttctca
R: tacagtttcatcccgatg
Отрицательный контроль - P.acid - HAMP
F: ttgaggtgtccgatgatg
R: ggaatgggagaagcagtt,
состав смеси ПЦР включает:
1. Дезоксинуклеотидтрифосфаты 2,5 мМ;
2. 10-кратный ПЦР Буфер;
3. MgCl2, 25 мМ;
4. Taq ДНК - полимераза с ингибирующими активность фермента антителами 5 Е/мкл;
5. Вода обработанная DEPC;
6. ЕvaGreen краситель 50x раствор в воде.
| WO 2017125893 A1, 27.07.2017 | |||
| WO 2017019440 А1, 02.02.2017 | |||
| БОГДАН Л.М | |||
| и др | |||
| Определение бактерий P | |||
| acnes на коже лица с помощью метода ПЦР-РВ, Youth for science 2020, Сборник статей II Международного учебно-исследовательского конкурса | |||
| Петрозаводск, 2020, стр | |||
| Счетный сектор | 1919 |
|
SU107A1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ НУКЛЕИНОВЫХ КИСЛОТ МЕТОДОМ ПОЛИМЕРАЗНО-ЦЕПНОЙ РЕАКЦИИ В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ И УСТРОЙСТВО ДЛЯ ЕГО ОСУЩЕСТВЛЕНИЯ | 2008 |
|
RU2385940C1 |
Авторы
Даты
2025-05-15—Публикация
2024-05-06—Подача