Изобретение относится к биотехнологии, генетической и белковой инженерии, и касается нового рекомбинантного штамма бактерий Escherichia coli (Е. coli), который может быть использован для получения рекомбинантного белка, представляющего собой химеру ДНК-зависимой ДНК-полимеразы семейства В из Thermococcus stetteri и ДНК-связывающего белка Sso7d из Sulfolbus solfataricus (TstP36H-Sso7d).
ДНК-полимеразы - это семейство ферментов, ответственных за точное дублирование ДНК. Полимеразы различаются по размеру, структуре, потребности в вспомогательных белках и роли в репликаций ДНК, также различаются и их специфические свойства, включая процессивность, точность синтеза, селективность к включаемым нуклеотидам. ДНК-полимеразы играют решающую роль не только в репликации и репарации ДНК in vivo, но и в методах, используемых в молекулярной биологии, особенно в полимеразной цепной реакции (ПЦР). ПЦР представляет собой быстрый, специфичный и чувствительный метод амплификации последовательностей нуклеиновых кислот, который широко используется в исследовательских и клинических лабораториях. Непрерывно ведется поиск новых ферментов для генетических технологий, которые могут иметь улучшенные свойства по сравнению с известными на сегодняшний день.
На данный момент в ПЦР активно используются ДНК-полимеразы, принадлежащие к семействам А и В. Наиболее известными представителями этих семейств являются полимеразы Taq из штамма бактерий Thermus aquaticus и Pfu из штамма бактерий Pyrococcus furiosus. С помощью методов генной инженерии для улучшения каталитических свойств были получены различные мутантные формы данных ферментов. Несмотря на многообещающие результаты, ограничениями для ПЦР являются выход продукта, длина ДНК, которая может быть амплифицирована, скорость полимеразы и точность процесса. В конечном счете, время выполнения ПЦР зависит исключительно от кинетических свойств фермента. Следует отметить, что не все коммерчески доступные ДНК-полимеразы обладают достаточной точностью и процессивностью. Так, частота ошибок Taq-полимеразы в 10 раз превышает таковую у Pfu при меньшем времени, требующемся для элонгации. ДНК-полимераза Pfu осуществляет элонгацию медленнее, но обладает 3'→5'-экзонуклеазной активностью, повышающей точность ДНК-полимеразы. Подобные ограничения при использовании известных в настоящее время ДНК-полимераз оставляют актуальной проблему поиска новых ферментов с улучшенными свойствами. Исследование малоизученных ДНК-полимераз позволяет выявить ферменты, потенциально обладающих свойствами, необходимыми для их использования для амплификации ДНК.
Гипертермофильные бактерии рода Thermococcus являются перспективным источником точных и термостабильных ДНК-полимераз. Thermococcus stetteri представляет собой гипертермофильную анаэробную архею, способную поддерживать жизнедеятельность в интервале температур от 55 до 94°С, что позволяет предположить, что ДНК-полимераза данного микроорганизма будет обладать высокой термостабильностью и в перспективе может использоваться в условиях ПЦР.
Известен штамм бактерий Thermococcus waiotapuensis - продуцент ДНК-полимеразы Twa (S. S. Cho. et al. Enzyme Microb Technol. 2012. 10;51(6-7):334-41. doi: 10.1016/j.enzmictec.2012.07.017). Для ДНК-полимеразы Twa из Thermococcus waiotapuensis показано, что фермент способен нарабатывать ампликоны до 6-8 kb со скоростью 1 kb/мин (S. S. Cho. et al. Enzyme Microb Technol. 2012. 10;51(6-7):334-41. doi: 10.1016/j.enzmictec.2012.07.017.).
Известен штамм бактерий Thermococcus barophilus Ch5 - продуцент ДНК-полимеразы Tba5 (K. - M. Kwon et al. Enzyme Microb Technol. 2016. 86:117-26. doi: 10.1016/j.enzmictec.2016.02.004). Для Tba5 из Thermococcus barophilus Ch5 показано, что фермент способен нарабатывать ампликоны до 10 kb со скоростью 1 kb/мин (K. - M. Kwon et al. Enzyme Microb Technol. 2016. 86:117-26. doi: 10.1016/j.enzmictec.2016.02.004).
Основным недостатком вышеназванных штаммов - продуцентов ДНК-полимераз, является низкая процессивность ферментов. В настоящее время скорость удлинения ДНК 1 kb/мин в условиях ПЦР считается низкой, например, коммерчески доступная ДНК-полимераза Phusion® High-Fidelity DNA Polymerase (New England Biolabs) производит удлинение ДНК со скоростью 2-4 kb/мин.
Наиболее близким к заявляемому штамму - прототипом, является штамм бактерий Escherichia coli - продуцент рекомбинантной ДНК-полимеразы Pfu из Pyrococcus furiosus (Заявка CN 114015672 А, опубл. 08.02.2022). Для данного фермента показана амплификация ДНК фрагментов длиной до 6 kb со скоростью 2,5 kb/мин.
Задачей изобретения является создание рекомбинантного штамма - продуцента химеры термостабильной ДНК-полимеразы семейства В из Thermococcus stetteri и ДНК-связывающего белка Sso7d из Sulfolbus solfataricus.
Технический результат изобретения заключается в получении рекомбинантного штамма-продуцента, обеспечивающего получение рекомбинантной химерной ДНК-полимеразы TstP36H-Sso7d с чистотой 95% и более высокой процессивностью фермента по сравнению с ферментом дикого типа.
Поставленная задача достигается тем, что на первом этапе с помощью векторной конструкции pET28c-Tst из штамма бактерий Thermococcus stetteri получают ДНК-полимеразу Tst дикого типа, способную эффективно нарабатывать фрагменты ДНК длиной до 6 kb за 2 мин (3 kb/мин). Для повышения процессивности фермента, на втором этапе, конструируют химеру Tst с ДНК-связывающим белком Sso7d, что позволяет нарабатывать фрагменты ДНК длиной до 10 kb за 2 мин (5 kb/мин). Поскольку фермент Дикого типа Tst, как и другие ДНК-полимеразы семейства В из архей, узнает урацил в ДНК матрице, что приводит к блокированию процесса синтеза ДНК, дополнительно была введена аминокислотная замена Р36Н, которая позволяет амплифицировать ДНК, содержащую остатки уридина (A. Tubeleviciute et al. Protein Eng Des Sel. 2010. 23(8):589-97. doi: 10.1093/protein/gzq032).
Заявляемый рекомбинантный штамм E.coli Rosetta 2(DE3)/pET28c-TstP36H-Sso7d-продуцент получают трансформацией культуры клеток Е. coli Rosetta 2(DE3) (генотип F- omp Thsd SB(rB-mB-) gal dcm (DE3) pRARE2 (CamR) рекомбинантной плазмидной ДНК pET28c-TstP36H-Sso7d, сконструированной на основе вектора рЕТ28 с, несущего ген, кодирующего ДНК-полимеразу В из Thermococcus stetteri, содержащую аминокислотную замену Р36Н и слитую с ДНК-связывающим белком Sso7d из Sulfolbus solfataricus.
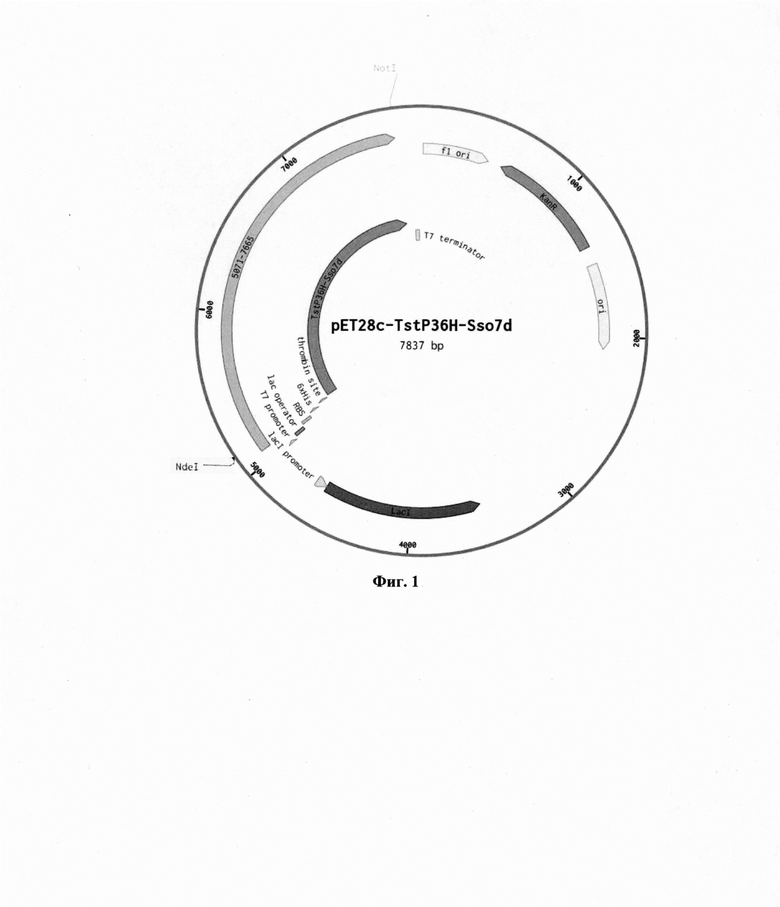
На фиг. 1 представлена физическая карта плазмиды pET28c-TstP36H-Sso7d, содержащей фрагмент ДНК, кодирующий ДНК-полимеразу В из Thermococcus stetteri с 1 по 775 аминокислотный остаток, содержащую аминокислотную замену Р36Н и слитую с ДНК-связывающим белком Sso7d из Sulfolbus solfataricus (69 аминокислотных остатков) и N-концевой гистидиновый тракт.Указаны сайты для эндонуклеаз рестрикции Not I, Nde I; сайт узнавания для тромбина; промотор РНК-полимеразы фага Т7; промотор lac I; фрагмент ДНК, кодирующий ДНК-полимеразу В из Thermococcus stetteri с 1 по 775 аминокислотный остаток, содержащую аминокислотную замену Р36Н и слитую с ДНК-связывающим белком Sso7d из Sulfolbus solfataricus (69 аминокислотных остатков) и N- концевой гистидиновый тракт; терминатор РНК-полимеразы фага Т7; ori репликации плазмиды, fl ori репликации для включения одноцепочечной репликации; KanR - ген устойчивости к канамицину.
Нуклеотидная последовательность рекомбинантной плазмиды pET28c-TstP36H-Sso7d (SEQ ID NO 1) и аминокислотная последовательность химерной ДНК-полимеразы TstP36H-Sso7d (SEQ ID NO 2) представлены в перечне последовательностей.
Культурально-морфологические особенности штамма Е. coli Rosetta 2(DE3)/pET28c-TstP36H-Sso7d: грамотрицательные прямые палочки, размером 1,1-1,5x2,0-3,0 мкм, одиночные, спор и капсул не образуют. Каталазоположительные. Оксидазоотрицательные. Факультативные анаэробы. Клетки хорошо растут на простых питательных средах, содержащих и не содержащих хлорамфеникол и канамицин, например, на среде LB (питательная среда Lisogeny Broth). На агаризованной среде - колонии гладкие, круглые, слабовыпуклые, с ровным краем. В жидких средах образуют равномерную светорассеивающую суспензию, при хранении без перемешивания оседают на дно. Клетки растут в интервале температур от 8°С до 43°С, интервал для наиболее эффективного культивирования - 28-38°С, оптимум роста при 37°С. Оптимальный интервал рН для культивирования рН 5-7. Изначальный штамм Е. coli Rosetta 2 (DE3) проявляет устойчивость к хлорамфениколу (34 мкг/мл), обусловленную наличием гена устойчивости в ДНК рекомбинантной плазмиды pRARE2.
Характеристики полезного вещества, синтезируемого штаммом: рекомбинантная химерная ДНК-полимераза TstP36H-Sso7d, слитая с ДНК-связывающим белком Sso7d из Sulfolbus solfataricus и несущая аминокислотную замену Р36Н, содержащая линкер (10 аминокислот) с сайтом узнавания тромбина и гистидиновую метку (6 аминоксилот).
Продуктивность созданного штамма рекомбинантной ДНК-полимеразы TstP36H-Sso7d составляет не менее 20% белка клеточного лизата при культивировании в жидкой питательной среде LB при 37 С, 250 об/мин, в условиях индукции 0,2 мМ ИПТГ (изопропил-р-0-1-тиогалактопиранозид) в течение 18 часов с добавлением антибиотика канамицина (50 мкг/мл).
Сущность изобретения поясняется следующими конкретными примерами получения и использования штамма Е. coli Rosetta 2(DE3)/pET28c-TstP36H-Sso7d.
Пример 1. Создание генетической конструкции, обеспечивающей синтез рекомбинантной химерной ДНК-полимеразы TstP36H-Sso7d в клетках Е. coli.
Из штамма клеток Thermococcus stetteri была выделена суммарная ДНК. На основании последовательности гена ДНК-полимеразы Tst, имеющейся в базе данных Uniprot, были синтезированы праймеры, соответствующие началу и концу гена и содержащие нуклеотидные последовательности с сайтами рестрикции для Nde I и Not I, Наработка гена ДНК-полимеразы Tst была произведена методом ПЦР.
Клонирование в экспрессионный вектор осуществлялось по сайтам рестрикции Nde I и Not I: фрагмент ДНК и вектор подвергались обработке обозначенными эндонуклеазами рестрикции, рестрикционные смеси очищались. Затем проводилось лигирование фрагментов при +4°С в течении 16 ч. После чего полученной лигазной смесью трансформировались клетки штамма Escherichia coli DH5a. Позитивные по данным метода ПЦР колонии были пересеяны в ночные культуры, из ночных культур выделялась плазмидная ДНК для подтверждения последовательности секвенированием по Сэнгеру. Последовательность нуклеотидов синтезированного фрагмента гена соотносится с необходимой аминокислотной последовательностью.
Далее методом сайт-направленного мутагенеза вводили аминокислотную замену Р36Н, а к С-концу ДНК-полимеразы Tst присоединили фрагмент гена ДНК-связывающего белка Sso7d из Sulfolbus solfataricus.
На фиг. 1 представлена физическая карта плазмиды pET28c-TstP36H-Sso7d, содержащая ориджин репликации fl, промотор РНК-полимеразы фага Т7, RBS-сайт посадки рибосомы, сайты рестрикции Nde I и Not I, по которым встроен фрагмент гена, кодирующего кодирующий ДНК-полимеразу TstP36H-Sso7d, гистидиновую метку 6×His (His-tag), терминатор РНК-полимеразы фага Т7, ген устойчивости к канамицину KanR, а также лактозный репрессор lad и лактозный оператор и промотор.
Полученная плазмида pET28c-TstP36H-Sso7d обеспечивает синтез в клетках Escherichia coli химерной ДНК-зависимой ДНК-полимеразы TstP36H-Sso7d, содержащей N-концевую гистидиновую метку, предназначенную для последующей очистки рекомбинантного белка TstP36H-Sso7d с помощью металлохелатной хроматографии.
Нуклеотидная последовательность рекомбинантной плазмиды pET28c-TstP36H-Sso7d (SEQ ID NO: 1) представлена в перечне последовательностей.
Пример 2. Получение штамма-продуцента рекомбинантной ДНК-полимеразы TstP36H-Sso7d и исследование его продуктивности.
Полученной плазмидой pET28c-TstP36H-Sso7d были трансформированы клетки Е. coli штамма Rosetta 2(DE3), содержащие в своем геноме ген, кодирующий ДНК- зависимую РНК-полимеразу фага Т7, под контролем промотора lacUV5, индуцируемого лактозой или ИПТГ. Кроме того, клетки Е. coli Rosetta 2(DE3) дефектны по генам протеаз Lon и ompT, что снижает деградацию гетерологичных белков.
В результате был получен штамм Е. coli Rosetta 2(DE3)/pET28c-TstP36H-Sso7d -продуцент рекомбинантной химерной ДНК-полимеразы TstP36H-Sso7d. Для поддержания полученного штамма - продуцента белка TstP36H-Sso7d использовали плотную агаризованную LB-среду, содержащую 50 мкг/мл канамицина.
Продуктивность полученного штамма-продуцента изучали путем культивирования клеток в среде LB, в термостатированном шейкере роторного типа при температуре 37°С, скорости вращения платформы 250 об/мин до оптической плотности А600=0,5-0,6. После этого индуцировали синтез целевого белка добавлением ИПТГ до конечной концентрации 0,2 мМ. В качестве контроля использовали культуру без добавления ИПТГ. Образцы собранной центрифугированием биомассы клеток Лизировали, используя пресс Френча, и анализировали методом электрофореза в 8%-ном полиакриламидном геле в системе Лэммли в денатурирующих условиях. В результате выявлено, что индукция 0,2 мМ ИПТГ культуры клеток Е. coli приводит к синтезу белка с молекулярной массой порядка 100 кДа, что соответствует ожидаемой молекулярной массе для рекомбинантного белка TstP36H-Sso7d с гистидиновой меткой (99,6 кДа). Количество рекомбинантного белка составило не менее 20% от клеточного лизата при культивировании в условиях индукции.
Пример 3. Очистка рекомбинантной химерной ДНК-полимеразы TstP36H-Sso7d и изучение ее активности.
Очистка ДНК-полимеразы TstP36H-Sso7d представляла собой трехэтапную методику. На первой стадии проводили металл-хелатную аффинную хроматографию с помощью сорбента Ni SepharoseTM High Performance, который связывает His-taq содержащие полипептиды. На второй стадии проводили дополнительную хроматографическую очистку на колонке Hi-Trap Heparin™, содержащей гепарин. Для третьей стадии очистки использовалась обращено-фазовая хроматография на колонке Hi-Trap Phenyl HP™. Фракции, содержащие TstP36H-Sso7d, объединяли, концентрировали на колонках Jet Biofil (Biofil, Китай). После этого добавили равный объем 100% глицерина, хранили полученный фермент при -20°С.
Результат трехстадийной очистки ДНК-полимеразы TstP36H-Sso7d представлен на фиг .2, где М - маркер подвижности 10-250 кДа (Kaleidoscope™); 1 - очищенная фракция рекомбинантной ДНК-полимеразы TstP36H-Sso7d.
Как видно на фиг. 2, препарат ДНК-полимеразы TstP36H-Sso7d является практически гомогенным, и его чистота составляет 95%.
Были определены оптимальные условия для протекания полимеразной реакции под действием ДНК-полимеразы Tst. Показано, что оптимальными буферными условиями являются: 20 мМ KCl, 3 мМ MgCl2 и 12,5 мМ (NH4)2SO4 при рН 8.4, оптимум температуры элонгации 72°С.
Эффективность ДНК-полимеразы TstP36H-Sso7d в процессе ПЦР была исследована на ДНК фага X с использованием праймеров, позволяющих получить ПЦР-продукты разной длины: 2 kb, 4 kb, 6 kb, 8 kb, 10 kb. В качестве фермента сравнения использовали фермент Tst дикого типа. Условия проведения ПЦР: предварительный прогрев смеси - 94°С в течении 4 мин; основной цикл - денатурация 94°С 10 с, отжиг праймеров 60°С 30 с, элонгация 72°С 2 мин; окончательная элонгация 72°С 10 мин; хранение 4°С ∞. Продукты реакции анализировали электрофорезом в 1,5%-агарозном геле в присутствии этидия бромистого с последующей визуализацией с помощью гель-документирующей системы Е-Вох СХ.5 TS (Vilber Lourman, France).
На фиг. 3 представлены результаты амплификации ДНК фрагментов. Видно, что ДНК-полимераза Tst дикого типа способна эффективно нарабатывать фрагменты ДНК длиной до 6 kb за 2 мин (3 kb/мин), в то время как для химерной ДНК-полимеразы TstP36H-Sso7d за это же время элонгации происходит эффективное накопление ампликонов размером до 10 kb (5 kb/мин), что свидетельствует об увеличении процессивности фермента TstP36H-Sso7d по сравнению с ферментом дикого типа.
Таким образом, создан штамм бактерий Escherichia coli Rosetta 2(DE3)/pET28c-TstP36H-Sso7d, являющийся продуцентом рекомбинантной химеры термостабильной ДНК-полимеразы семейства В из Thermococcus stetteri и ДНК-связывающего белка Sso7d из Sulfolbus solfataricus и способной амплифицировать ДНК в условиях ПЦР.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3"
fileName="pET28c-TstP36H-Sso7d.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2024-07-18">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2024-07-18</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2024-07-18</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт химической биологии и
фундаментальной медицины Сибирского отделения Российской академии
наук (ИХБФМ СО РАН)</ApplicantName>
<ApplicantNameLatin>The Institute of Chemical Biology and
Fundamental Medicine of the Siberian Branch of the Russian Academy of
Sciences</ApplicantNameLatin>
<InventorName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт химической биологии и
фундаментальной медицины Сибирского отделения Российской академии
наук (ИХБФМ СО РАН)</InventorName>
<InventorNameLatin>The Institute of Chemical Biology and Fundamental
Medicine of the Siberian Branch of the Russian Academy of
Sciences</InventorNameLatin>
<InventionTitle languageCode="ru">Рекомбинантный штамм бактерий
Escherichia coli Rosetta 2(DE3)/pET28с-TstP36H-Sso7d – продуцент
химеры термостабильной ДНК-полимеразы семейства B из Thermococcus
stetteri и ДНК-связывающего белка Sso7d из Sulfolbus
solfataricus</InventionTitle>
<SequenceTotalQuantity>2</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>7837</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7837</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggcgaatgggacgcgccctgtagcggcgcattaagcgcggcgggtgtg
gtggttacgcgcagcgtgaccgctacacttgccagcgccctagcgcccgctcctttcgctttcttccctt
cctttctcgccacgttcgccggctttccccgtcaagctctaaatcgggggctccctttagggttccgatt
tagtgctttacggcacctcgaccccaaaaaacttgattagggtgatggttcacgtagtgggccatcgccc
tgatagacggtttttcgccctttgacgttggagtccacgttctttaatagtggactcttgttccaaactg
gaacaacactcaaccctatctcggtctattcttttgatttataagggattttgccgatttcggcctattg
gttaaaaaatgagctgatttaacaaaaatttaacgcgaattttaacaaaatattaacgtttacaatttca
ggtggcacttttcggggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgt
atccgctcatgaattaattcttagaaaaactcatcgagcatcaaatgaaactgcaatttattcatatcag
gattatcaataccatatttttgaaaaagccgtttctgtaatgaaggagaaaactcaccgaggcagttcca
taggatggcaagatcctggtatcggtctgcgattccgactcgtccaacatcaatacaacctattaatttc
ccctcgtcaaaaataaggttatcaagtgagaaatcaccatgagtgacgactgaatccggtgagaatggca
aaagtttatgcatttctttccagacttgttcaacaggccagccattacgctcgtcatcaaaatcactcgc
atcaaccaaaccgttattcattcgtgattgcgcctgagcgagacgaaatacgcgatcgctgttaaaagga
caattacaaacaggaatcgaatgcaaccggcgcaggaacactgccagcgcatcaacaatattttcacctg
aatcaggatattcttctaatacctggaatgctgttttcccggggatcgcagtggtgagtaaccatgcatc
atcaggagtacggataaaatgcttgatggtcggaagaggcataaattccgtcagccagtttagtctgacc
atctcatctgtaacatcattggcaacgctacctttgccatgtttcagaaacaactctggcgcatcgggct
tcccatacaatcgatagattgtcgcacctgattgcccgacattatcgcgagcccatttatacccatataa
atcagcatccatgttggaatttaatcgcggcctagagcaagacgtttcccgttgaatatggctcataaca
ccccttgtattactgtttatgtaagcagacagttttattgttcatgaccaaaatcccttaacgtgagttt
tcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcg
taatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctacc
aactctttttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtgtagccg
tagttaggccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccag
tggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggc
gcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactg
agatacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccgg
taagcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatctttatag
tcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagccta
tggaaaaacgccagcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttct
ttcctgcgttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgataccgctcgccg
cagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgcctgatgcggtattttctc
cttacgcatctgtgcggtatttcacaccgcatatatggtgcactctcagtacaatctgctctgatgccgc
atagttaagccagtatacactccgctatcgctacgtgactgggtcatggctgcgccccgacacccgccaa
cacccgctgacgcgccctgacgggcttgtctgctcccggcatccgcttacagacaagctgtgaccgtctc
cgggagctgcatgtgtcagaggttttcaccgtcatcaccgaaacgcgcgaggcagctgcggtaaagctca
tcagcgtggtcgtgaagcgattcacagatgtctgcctgttcatccgcgtccagctcgttgagtttctcca
gaagcgttaatgtctggcttctgataaagcgggccatgttaagggcggttttttcctgtttggtcactga
tgcctccgtgtaagggggatttctgttcatgggggtaatgataccgatgaaacgagagaggatgctcacg
atacgggttactgatgatgaacatgcccggttactggaacgttgtgagggtaaacaactggcggtatgga
tgcggcgggaccagagaaaaatcactcagggtcaatgccagcgcttcgttaatacagatgtaggtgttcc
acagggtagccagcagcatcctgcgatgcagatccggaacataatggtgcagggcgctgacttccgcgtt
tccagactttacgaaacacggaaaccgaagaccattcatgttgttgctcaggtcgcagacgttttgcagc
agcagtcgcttcacgttcgctcgcgtatcggtgattcattctgctaaccagtaaggcaaccccgccagcc
tagccgggtcctcaacgacaggagcacgatcatgcgcacccgtggggccgccatgccggcgataatggcc
tgcttctcgccgaaacgtttggtggcgggaccagtgacgaaggcttgagcgagggcgtgcaagattccga
ataccgcaagcgacaggccgatcatcgtcgcgctccagcgaaagcggtcctcgccgaaaatgacccagag
cgctgccggcacctgtcctacgagttgcatgataaagaagacagtcataagtgcggcgacgatagtcatg
ccccgcgcccaccggaaggagctgactgggttgaaggctctcaagggcatcggtcgagatcccggtgcct
aatgagtgagctaacttacattaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtg
ccagctgcattaatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgccagggtggtttt
tcttttcaccagtgagacgggcaacagctgattgcccttcaccgcctggccctgagagagttgcagcaag
cggtccacgctggtttgccccagcaggcgaaaatcctgtttgatggtggttaacggcgggatataacatg
agctgtcttcggtatcgtcgtatcccactaccgagatatccgcaccaacgcgcagcccggactcggtaat
ggcgcgcattgcgcccagcgccatctgatcgttggcaaccagcatcgcagtgggaacgatgccctcattc
agcatttgcatggtttgttgaaaaccggacatggcactccagtcgccttcccgttccgctatcggctgaa
tttgattgcgagtgagatatttatgccagccagccagacgcagacgcgccgagacagaacttaatgggcc
cgctaacagcgcgatttgctggtgacccaatgcgaccagatgctccacgcccagtcgcgtaccgtcttca
tgggagaaaataatactgttgatgggtgtctggtcagagacatcaagaaataacgccggaacattagtgc
aggcagcttccacagcaatggcatcctggtcatccagcggatagttaatgatcagcccactgacgcgttg
cgcgagaagattgtgcaccgccgctttacaggcttcgacgccgcttcgttctaccatcgacaccaccacg
ctggcacccagttgatcggcgcgagatttaatcgccgcgacaatttgcgacggcgcgtgcagggccagac
tggaggtggcaacgccaatcagcaacgactgtttgcccgccagttgttgtgccacgcggttgggaatgta
attcagctccgccatcgccgcttccactttttcccgcgttttcgcagaaacgtggctggcctggttcacc
acgcgggaaacggtctgataagagacaccggcatactctgcgacatcgtataacgttactggtttcacat
tcaccaccctgaattgactctcttccgggcgctatcatgccataccgcgaaaggttttgcgccattcgat
ggtgtccgggatctcgacgctctcccttatgcgactcctgcattaggaagcagcccagtagtaggttgag
gccgttgagcaccgccgccgcaaggaatggtgcatgcaaggagatggcgcccaacagtcccccggccacg
gggcctgccaccatacccacgccgaaacaagcgctcatgagcccgaagtggcgagcccgatcttccccat
cggtgatgtcggcgatataggcgccagcaaccgcacctgtggcgccggtgatgccggccacgatgcgtcc
ggcgtagaggatcgagatctcgatcccgcgaaattaatacgactcactataggggaattgtgagcggata
acaattcccctctagaaataattttgtttaactttaagaaggagatataccatgggcagcagccatcatc
atcatcatcacagcagcggcctggtgccgcgcggcagccatatgatcctcgacacagactacataaccga
ggatggaaaacccgttatcaggatcttcaagaaggagaacggcgagttcaagattgactacgataggact
ttcgaacattacttctacgcccttctgaaggacgactctgctattgaagacgttaagaagataaccgctg
agaggcgcggaacggttgtaactgtaaagtgcgctgagaaagtggagaagaagttccttggaagaccggt
tgaagtgtggaagctctactttacccacccacaggacgtcccggcaatccgcgataaaatacggaagcat
ccagcagttattgatatctacgagtacgacatacccttcgccaagcgctacctaatcgacaagggcctga
tcccgatggagggtgacgaggagctgaagatgctcgccttcgatatcgaaacgctctaccacgagggcga
ggagttcgccgaagggcccattctgatgataagctacgctgacgaggaaggggcgagggtaataacgtgg
aagaacgtagaccttccatacgttgacgtcgtctcgacggaaagggagatgataaagcgctttctcaggg
tcgtcaaggagaaagaccccgacatactgataacctacaacggcgacaacttcgacttcgcctatctgaa
gaagcgctgtgaaaagctcggaataaacttcgcgctcggaagggacggaagcgagccgaagatccagaga
atgggcgaccgcttcgccgttgaggtgaaggggcggatacactttgacctctaccctgtgataagaagga
cgataaacctgccgacctacaaccttgaagccgtctacgaggccatctttggaaagccgaaggagaaggt
ctacgccgaggagatagcgcaggcatgggagagcggcgaggggcttgaaagggtagctcagtactcgatg
gaggacgccaaagtaacctacgagctcggaaaggagttcttcccgatggaagcccagctttcccgcttga
tcggccagtccctctgggacgtctcccgctcaagcacgggaaacctcgtcgagtggttccttcttaggaa
ggcctacgagaggaacgagctggccccgaacaagcccgatgaaagagaacttgcaaagagaagggaaagc
tacaccggcggatacgtcaaagagcctgagagagggctctgggagaacatcgtctatctggatttccgct
cgctgtacccatcgataataatcacccacaacgtctctccagatactctcaacagagaaggatgcaggga
gtatgatatcgctccacaggtgggccacaggttctgcaaggacttcccgggcttcatccctagcctgctg
ggagacctgcttgaggagaggcagaagataaagaaaaggatgaaggccacgattgacccgatcgagaaga
aaattctcgattacaggcagagggcaattaagatcctcgcaaacagcttctacggctactacggctatgc
caaagcacggtggtactgcagggagtgcgcggagagcgtcaccgcctgggggagggagtacataacgatg
acgatcagagagatagagaagaagtacggctttaaagtgctgtacgcagacactgacggcttcttcgcga
caataccaggatctgacgccgaaaccgtcaaaaagaaggcaatggagttcctcaaatacatcaacgacaa
acttccgggtgcgctcgagctcgagtacgaggggttctacaggcgcggcttctttgtgacgaagaaaaag
tacgcggtcatcgacgaggagggcaagataacgacgcgcggacttgagatagtcagacgcgattggagcg
agatagcaaaggagacgcaggcgagggttcttgaagcgttgctgaaggacggtgacgttgagaaggccgt
cagcatagtcaaggaagtgacagagaagctgagcaagtacgaggttccgccggagaagctggtgatccac
gagcagataacgagggatctgaggcactacaaagcaaccggcccgcacgttgccgtggctaagaggctgg
cggcgaggggagtaaagataagaccgggaactgtcatcagctacatcgttctcaaaggctctggaaggat
aggtgacagggcgataccgttcgacgagttcgacccgacgaagcacaagtacgacgccgattactacatc
gagaaccaggttctgccggccgttgagagaatcctaaaggcccttggctaccgggcagaagatttgcgct
accagaagacgaggcaggttggtttgggtgcctggctgaagccgaaggggaaggggggtaccggcggtgg
cggtgcaaccgtaaagttcaagtacaaaggcgaagaaaaagaggtagacatctccaagatcaagaaagta
tggcgtgtgggcaagatgatctccttcacctacgacgagggcggtggcaagaccggccgtggtgcggtaa
gcgaaaaggacgcgccgaaggagctgctgcagatgctggagaagcagaaaaagtgagcggccgcactcga
gcaccaccaccaccaccactgagatccggctgctaacaaagcccgaaaggaagctgagttggctgctgcc
accgctgagcaataactagcataaccccttggggcctctaaacgggtcttgaggggttttttgctgaaag
gaggaactatatccggat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>864</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..864</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MGSSHHHHHHSSGLVPRGSHMILDTDYITEDGKPVIRIFKKENGEFKID
YDRTFEHYFYALLKDDSAIEDVKKITAERRGTVVTVKCAEKVEKKFLGRPVEVWKLYFTHPQDVPAIRDK
IRKHPAVIDIYEYDIPFAKRYLIDKGLIPMEGDEELKMLAFDIETLYHEGEEFAEGPILMISYADEEGAR
VITWKNVDLPYVDVVSTEREMIKRFLRVVKEKDPDILITYNGDNFDFAYLKKRCEKLGINFALGRDGSEP
KIQRMGDRFAVEVKGRIHFDLYPVIRRTINLPTYNLEAVYEAIFGKPKEKVYAEEIAQAWESGEGLERVA
QYSMEDAKVTYELGKEFFPMEAQLSRLIGQSLWDVSRSSTGNLVEWFLLRKAYERNELAPNKPDERELAK
RRESYTGGYVKEPERGLWENIVYLDFRSLYPSIIITHNVSPDTLNREGCREYDIAPQVGHRFCKDFPGFI
PSLLGDLLEERQKIKKRMKATIDPIEKKILDYRQRAIKILANSFYGYYGYAKARWYCRECAESVTAWGRE
YITMTIREIEKKYGFKVLYADTDGFFATIPGSDAETVKKKAMEFLKYINDKLPGALELEYEGFYRRGFFV
TKKKYAVIDEEGKITTRGLEIVRRDWSEIAKETQARVLEALLKDGDVEKAVSIVKEVTEKLSKYEVPPEK
LVIHEQITRDLRHYKATGPHVAVAKRLAARGVKIRPGTVISYIVLKGSGRIGDRAIPFDEFDPTKHKYDA
DYYIENQVLPAVERILKALGYRAEDLRYQKTRQVGLGAWLKPKGKGGTGGGGATVKFKYKGEEKEVDISK
IKKVWRVGKMISFTYDEGGGKTGRGAVSEKDAPKELLQMLEKQKK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---

Изобретение относится к биотехнологии, генетической и белковой инженерии и представляет собой рекомбинантный штамм бактерий Escherichia coli Rosetta 2(DE3)/pET28c-TstP36H-Sso7d - продуцент химерной термостабильной ДНК-полимеразы TstP36H-Sso7d, содержащей ДНК-зависимую ДНК-полимеразу семейства В из Thermococcus stetteri с аминокислотной заменой Р36Н, и ДНК-связывающий белок Sso7d из Sulfolbus solfataricus, полученный путем трансформации клеток штамма Escherichia coli Rosetta 2(DE3) плазмидой ДНК pET28c-TstP36H-Sso7d, сконструированной на основе вектора рЕТ28с, и имеющей нуклеотидную последовательность SEQ ID NO: 1, содержащую ген, кодирующий ДНК-полимеразу TstP36H-Sso7d, имеющую аминокислотную последовательность SEQ ID NO: 2. Изобретение позволяет получить рекомбинантный штамм-продуцент, обеспечивающий получение рекомбинантной ДНК-полимеразы TstP36H-Sso7d с чистотой 95% и с более высокой процессивностью по сравнению с ферментом дикого типа. 3 ил., 3 пр.
Рекомбинантный штамм бактерий Escherichia coli Rosetta 2(DE3)/pET28c-TstP36H-Sso7d - продуцент химерной термостабильной ДНК-полимеразы TstP36H-Sso7d, содержащей ДНК-зависимую ДНК-полимеразу семейства В из Thermococcus stetteri с аминокислотной заменой Р36Н, и ДНК-связывающий белок Sso7d из Sulfolbus solfataricus, полученный путем трансформации клеток штамма Escherichia coli Rosetta 2(DE3) плазмидой ДНК pET28c-TstP36H-Sso7d, сконструированной на основе вектора рЕТ28с, и имеющей нуклеотидную последовательность SEQ ID NO: 1, содержащую ген, кодирующий ДНК-полимеразу TstP36H-Sso7d, имеющую аминокислотную последовательность SEQ ID NO: 2.
| CN114015672, 08.02.2022 | |||
| Машина для производства земляных работ | 1932 |
|
SU29152A1 |
| ТЕРМОСТАБИЛЬНАЯ ДНК-ПОЛИМЕРАЗА, СПОСОБ ЕЕ ПОЛУЧЕНИЯ, ФРАГМЕНТ ВЫДЕЛЕННОЙ ДНК, СПОСОБ АМПЛИФИКАЦИИ ДНК, СПОСОБ ВВЕДЕНИЯ МЕТКИ В ДНК И СПОСОБ ОБРАТНОЙ ТРАНСКРИПЦИИ | 1997 |
|
RU2197524C2 |
| МОДИФИЦИРОВАННАЯ ТЕРМОСТАБИЛЬНАЯ ДНК-ПОЛИМЕРАЗА, СПОСОБ ЕЁ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЕ | 1997 |
|
RU2235773C2 |
| WO 1998014590 A1, 09.04.1998 | |||
| ПРИСПОСОБЛЕНИЕ ДЛЯ НАМЕТКИ ПЕТЕЛЬ И ПУГОВИЦ ПРИ ПОШИВКЕ ОДЕЖДЫ | 1932 |
|
SU33100A1 |
Авторы
Даты
2025-05-19—Публикация
2024-07-26—Подача