Техническая часть
Изобретение касается материалов и методологий, которыми руководствуются в случаях инфекции вирусом не-A, не-B гепатита (NANBV). Более подробно, она относится к этиологическому агенту не-A, не-B гепатита (NANBH), вирусу гепатита C (HCV) и к полинуклеотидам и их аналогам, которые используются для определения HCV в биологических образцах.
Основания для изобретения
Не-A, не-B гепатит (NANBH) - передающаяся от одного человека другому болезнь или семейство заболеваний, которая, как считают, вирусного происхождения, и неотличима от других форм вирус-ассоциированных болезней печени, включая и те, что вызываются известными вирусами гепатита, т.е. вирусом гепатита A (HAV), гепатита B (HBV) и вирусом гепатита дельта (HDV) так же, как и гепатит, вызываемый цитомегаловирусом (CMV) или вирусом Эпштейн-Бара (EBV). NANBH был сначала выявлен у индивидуумов, которым сделали переливание крови. Передача от человека шимпанзе и серийный пассаж в шимпанзе обнаружил свидетельство о том, что NANBH обусловливается заразным инфекционным агентом или агентами.
Эпидемиологическое доказательство предполагает, что возможны три типа NANBH: передающийся посредством воды эпидемический тип, через кровь или иглы и спорадически возникающий (заражение в обществе других людей) тип. Однако количество агентов, которые могут вызывать NANBH, неизвестно.
Есть целый ряд кандидатов в NANBH. См., например, обзоры Prince (1983), Feinstone и Hoofnagle (1984), Overby (1985, 1986, 1987) и статья Iwarson (1987). Однако нет доказательства, что какой-либо из этих кандидатов представляет этиологический агент NANBH.
Потребность в чувствительных специфических методах для скрининга и идентификации носителей NANBV и NANBV, зараженной крови или ее компонентов, очень важна. Посттрансфузионный гепатит (PTH) случается приблизительно у 10% пациентов, и NANBH отвечает за максимум 90% этих случаев. Основная проблема этой болезни - частое прогрессирование к хроническому повреждению печени (25-55%).
Обслуживание пациентов, как и предотвращение заражения NANBH через кровь и ее компоненты или путем тесных персональных контактов, требует надежных инструментов скрининга, диагностики и прогностики для обнаружения нуклеиновых кислот, антигенов и антител, относящихся NANBH.
Методы обнаружения специфических полинуклеотидов гибридизационными анализами известны в науке. См., например, Matthews и Kricka (1988), Analytical Biochemistry 169: 1; Landegren и др. (1988), Science 242:229 и Mittlin (1989), Clinical chem 35:1819, US patent N 4868105, выданный Sept., 9, 1989, и в EPO publication N 225807 (опубликованном 16 июня, 1987).
Заявитель обнаружил новый вирус, вирус гепатита C (HCV), который, как доказали, является главным этиологическим агентом передающегося посредством воды NANBH (BB-NANBH). Первая работа заявителя, включающая частичную геномную последовательность изолята прототипа HCV, CDC /HCV1/, также обозначенного как HCV1, описана в EPO публикации N 318216 PCT N WO 89/04669 (опубликованной 31 мая 1989 г.) и опубликованного 1 июня 1989 г. Эти патентные заявления так же, как любые соответствующие национальные патентные заявления, объединены здесь в сносках. Эти заявления учат методам рекомбинантной ДНК - клонирования последовательностей HCV, диагностическим процедурам с пробой HCV, антиHCV-антител и методам выделения новых последовательностей.
Раскрытие изобретения
Настоящее изобретение основано на последовательностях HCV, описанных EPO publication N 318216 и PCT pub. N WO 89/04669, так же, как и на других последовательностях HCV, которые описаны здесь. Методы выделения и/или детекции специфических полинуклеотидов гибридизацией не могло быть использовано для скрининга HCV до открытия заявителем HCV. Соответственно один из аспектов изобретения - олигомер, способный гибридизоваться с последовательностью HCV в аналит-полинуклеотидной цепи, где олигомер включен в последовательность-мишень HCV, комплементарную по крайней мере 4 прилежащим нуклеотидам кДНК HCV, показанной на фиг. 18.
Другой аспект изобретения - процесс обнаружения последовательности HCV в цепи аналита, подозрительного на содержение HCV полинуклеотида, где HCV полинуклеотид включает селективный район мишени, названный процесс включает:
а) обеспечение олигомером, способным гибридизоваться с HCV последовательностью в аналит-полинуклеотидной цепи, где олигомер включает HCV последовательность-мишень, комплементарную по крайней мере 4 прилежащим нуклеотидам HCV кДНК, представленной на фиг. 18,
б) инкубацию цепи аналита с олигомером а), что позволяет формировать специфические гибридные дуплексы между последовательностью мишени и исследуемой последовательностью и
в) детекцию гибридов, образованных между районом мишени или любым другим и олигомером.
Еще один аспект изобретения - метод получения крови, свободной от HCV, включает:
а) обеспечение аналит-нуклеиновыми кислотами из образцов крови, подозрительных на содержание HCV последовательности-мишени,
б) обеспечение олигомером, способным гибридизоваться с последовательностью HCV в аналит-полинуклеотидной цепи или любой другой, где олигомер включает HCV последовательность мишени, комплементарную последовательности по крайней мере 8 нуклеотидов, присутствующих в консервативной нуклеотидной последовательности HCV в PHK,
в) реагирование а) с б) при условиях, которые позволяют образовать полинуклеотидный дуплекс между последовательностью мишени и исследуемой последовательностью, если не любой другой,
г) обнаружение сформированного дуплекса в в) или любого другого и
д) сохранение крови, в которой не обнаруживались комплексы в г).
Краткое описание чертежей
Фиг. 1 показывает последовательность HCV кДНК в клоне 12f и кодируемую им аминокислоту.
Фиг. 2 демонстрирует последовательность кДНК HCV в клоне K9-1 и кодируемые им аминокислоты.
Фиг. 3 демонстрирует последовательность клона 15e и кодируемые им аминокислоты.
Фиг. 4 представляет нуклеотидную последовательность кДНК HCV в клоне 13i, кодируемые им аминокислоты и последовательности, которые перекрываются клоном 12f.
Фиг. 5 демонстрирует нуклеотидную последовательность кДНК HCV в клоне 26j, кодируемые им аминокислоты и последовательности, которые перекрывают клон 13i.
Фиг. 6 показывает нуклеотидную последовательность кДНК HCV в клоне CA59a, кодируемые им аминокислоты и последовательности, которые перекрываются с клонами 26j и K9-1.
Фиг. 7 - 17 демонстрируют нуклеотидную HCV кДНК, которая перекрывается соответственно:
на фиг. 7 в клоне CA84a клоном CA59a;
на фиг. 8 в клоне CA156e клоном CA84a;
на фиг. 9 в клоне CA1676 клоном CA156e;
на фиг. 10 в клоне CA216a клоном CA1676;
на фиг. 11 в клоне CA290a клоном CA216a;
на фиг. 12 в клоне ад30a клоном CA290a;
на фиг. 13 в клоне CA205a последовательностью HCV кДНК в клоне CA290a;
на фиг. 14 в клоне 18д последовательностью HCV кДНК в клоне ag30a;
на фиг. 15 в клоне 16jh последовательностью HCV кДНК в клоне 15e;
на фиг. 16 в клоне 6k последовательностью HCV кДНК в клоне 16jh;
на фиг. 17 в клоне p131jh последовательностью HCV кДНК в клоне 6к.
Фиг. 18 представляет составленную кДНК последовательность HCV, происходящую из клонов, здесь описанных, и их составной HCV кДНК последовательности, представленной в БРО публикации N 318216. Клонами, из которых произошли эти последовательности, являются
5'-clone 32, b114a, 18g, ag30a, CA205a, CA290a, CA216a, pi14a, CA167b, CA156e, CA84a, CA59a, K9-1, 26j, 13i, 12f, 14i, 11b, 7f, 7e, 8h, 33c, 40b, 37b, 35, 36, 81, 32, 33b, 25c, 14c, 8f, 33f, 33g, 39c, 35f, 19g, 26g, 15e, b5a, 16jh, 6k и p131jh. На чертеже три горизонтальных штриха выше последовательности указывают положение предположительного инициаторного метионинового кодона. Также на чертеже показана аминокислотная последовательность предположительного полипротеина, кодируемого кДНК HCV. Гетерогенность в клонированных ДНК HCV1 указана аминокислотами, приведенными выше предположительной кодирующей последовательности большой OPC, круглые скобки указывают, что гетерогенность обнаружена в или около 5'- или 3'-конца HCV кДНК клона.
Фиг. 19 представляет последовательности захваченных и меченых проб для детекции PHK HCV в биологических образцах.
Фиг. 20 показывает схематический вид полипротеина флавивируса и предположительного полипротеина HCV, кодируемого в большой OPC генома HCV. Также на чертеже указаны возможные функции полипептидов флавивируса, очищенных из флавивирусных полипротеинов. В дополнение, приведены сравнительные расположения полипептидов NANB 5-1-1 и C100 по сравнению с предположительным HCV полипротеином.
Фиг. 21 показывает двухцепочечную нуклеотидную последовательность инсерции кДНК HCV в клоне и предположительной аминокислотной последовательности кодируемого ею полипептида.
Фиг. 22 дает HCV кДНК последовательность в клоне 36, сегмент которой перекрывается с кДНК NANBV клона 81, и кодируемую клоном 36 полипептидную последовательность.
Фиг. 23 дает кДНК HCV последовательность в клоне 37b, сегмент которой перекрывается с клоном 35, и кодируемый им полипептид.
Фиг. 24 дает авторадиографии HCV кПЦР анализа на РНК, полученной из образцов печени шимпанзе с NANBH (фиг. 25A) и итальянских пациентов с NANBH (фиг. 25B).
Фиг. 25A и 25B - графики, показывающие температурную зависимость между степенью повреждения печени, присутствием HCV РНК и присутствием антиHCV-антител для двух шимпанзе с NANBH.
Фиг. 26 дает нуклеотидную последовательность HCV кДНК в клоне CAB4a, кодируемые ею аминокислоты и последовательности, перекрывающиеся с клоном C459a.
Фиг. 27 дает HCV кДНК последовательность в клоне 40b, сегмент которой перекрывается с клоном 37b, и кодируемый ею полипептид.
Фиг. 28 - авторадиография, показывающая меченые амплифицированные продукты приблизительно 300, 30 и 3 CID HCV геномов.
Фиг. 29 дает нуклеотидную последовательность HCV кДНК в клоне 40a.
Фиг. 30 - авторадиография, показывающая амплифицированные продукты, полученные с праймеров, происходящих из консервативных районов HCV генома.
Фиг. 31 представляет HCV кДНК последовательность в клоне 35, сегмент которого перекрывает клон 36, и кодируемый ею полипептид.
Фиг. 32 - диаграмма, показывающая связь проб и праймеров, происходящих из 5'-района HCV РНК, из которого происходят HCV кДНК клонов ag30a и K9-1.
Фиг. 33 - авторадиография амплифицированных продуктов, полученных с наборов праймеров, происходящих от ag30a и K9-1.
Фиг. 34 дает построчно нуклеотидные последовательности человеческих изолятов 23 и 27 HCV 1. Гомологичные последовательности указаны символом (*). Негомологичные последовательности в маленьком шрифте.
Фиг. 35 демонстирует построчно аминокислотные последовательности изолятов человека 23 и 27 HCV 1. Гомологичные последовательности указаны символом (*). Негомологичные последовательности - маленьким шрифтом.
Фиг. 36 показывает изображение в полутонах радиоавтографа норзерн блота РНК, изолированной из печи инфицированной BB-NANBV шимпанзе, гибридизации с BB-NANBV кДНК клона 81.
Фиг. 37 дает изображение в полутонах радиоавтографа нуклеиновых кислот, экстрагированных из NANBV частиц, захваченных из инфицированной плазмы с помощью антиNANB 5-1-1 и гибридизованных с 32P-меченой NANBV кДНК из клона 81.
Фиг. 38A и B дает изображение радиоавтографов фильтров, содержащих выделенные NANBV нуклеиновые кислоты, гибридизованные с 32P-мечеными плюс- и минус-цепями ДНК в качестве проб, которые происходили из NANBV кДНК в клоне 81.
Фиг. 39 демонстрирует нуклеотидную последовательность консенсуса изолята человека 23, вариантные последовательности показаны ниже строки этой последовательности. Также показаны аминокислоты, кодируемые последовательностью консенсуса.
Фиг. 40 - график, показывающей связь праймеров EnvR и EnvL с моделью полипротеина флавивируса и предположительно HCV полипротеина.
Фиг. 41 дает сравнение (соответствие по вертикали) нуклеотидных последовательностей изолятов Thorn, ECI, HCT # 18 и HCV 1.
Фиг. 42 дает сравнение нуклеотидных последовательностей EC10 и составной HCV1 последовательностью, последовательность EC10 расположена в строчке выше дотов, и последовательность в строчке ниже дотов.
Фиг. 43 дает сравнение аминокислотных последовательностей 117-308 (относительно HCV1), кодируемых в "EnvL" районах последовательностей консенсуса изолятов человека HCT # 18, JH23, JH27, Thorne, EC1 и HCV 1.
Фиг. 44 дает сравнение аминокислотных последовательностей 330-360 (относительно HCV1), кодируемых в "EnvR" районах последовательностей консенсуса изолятов человека HCT # 18, JH23, JH27, Thorne, EC1 и HCV1.
Фиг. 45 дает нуклеотидные последовательности индивидуальных праймеров в праймерной смеси 5'-3.
Способы осуществления изобретения
Термин "вирус гепатита C" (HCV) заранее заказан исследователями этой области для до сих пор неизвестного этиологического агента NANBH. Прототипный изолят HCV был идентифицирован USSN 122, 714 (см. также EPO publication N 318,216). Термин HCV также включает новые изоляты тех же вирусных видов. Как разъяснение этой терминологии заболевание, вызываемое HCV, ранее названное передающийся посредством воды NANB гепатит (BB-NANBH), назвали гепатитом C. Термины NANBH и гепатит C могут использоваться в этом случае взаимозаменяемо.
HCV-вирусные виды, посредством которых патогенные штаммы вызывают BB-NANBH. Могут быть также ослабленные штаммы или дефектные мешающие частицы, берущие от них начало. Как показано ниже, HCV геном состоит из РНК. Известно, что РНК-содержащие вирусы имеют относительно высокие скорости спонтанных мутаций, т.е. явно порядка 10-3 - 10-4 на включившийся нуклеотид (Eields и Knipe (1986)). Следовательно, так как гетерогенность и "текучесть" генотипа наследуется в РНК-вирусах, среди их штаммов есть множество штаммов (изолятов), которые могут быть вирулентными или авирулентными. Рецептуры и методы, описанные здесь, дают возможность распространять, идентифицировать, обнаруживать и выделять различные HCV штаммы или изоляты.
Идентифицировано несколько разных штаммов/изолятов HCV (см. ниже). Один такой штамм или изолят, который является прототипом, обозначен CDC/HCV1 (также названный HCV1). Информации об одном штамме или изоляте, таком как частичная геномная последовательность, достаточно, чтобы позволить тем, кто квалифицирован в этой области, используя стандартные технологии, выделить новые штаммы/изоляты и идентифицировать, являются ли также новые штаммы/изоляты HCV. Например, несколько разных штаммов/изолятов описано ниже. Эти штаммы, которые были получены из целого ряда человеческих сывороток (и из разных географических областей), изолировали, используя информацию о геномной последовательности HCV1.
Используя технологии, описанные в EPO publication N 318,216 и ниже, была определена геномная структура и нуклеотидная последовательность HCV1 геномной РНК. Оказалось, что геном - одноцепочечная РНК, содержащая ≈ 10 тыс. нуклеотидов. Геном - позитивная цепь и обладает продолжительной трансляционной открытой рамкой считывания (OPC), которая кодирует полибелок около 3000 аминокислот. По-видимому, в OPC структурные белки кодируются приблизительно первой четвертью N-терминального района, причем большая часть полибелка ответственна за неструктурные белки. При сравнении со всеми известными вирусными последовательностями наблюдаются маленькие, но важные гомологии с неструктурными белками семейства флавивирусов и с пестивирусами (которые теперь также рассматривают как часть семейства флавивирусов).
Схематическое выравнивание возможных районов флавивирусного полибелка (используя в качестве примера вирус желтой лихорадки) и предположительно полибелка, закодированного в главной OPC генома HCV, показано на фиг. 20. На чертеже указаны возможные домены HCV полибелка. Полибелок флавивируса содержит, с амино конца к С-терминальному концу, белок нуклеокапсида (C), белок матрикса (M), белок оболочки (E), неструктурные белки (NS) 1, 2 (a + b), 3, 4 (a + b) и 5. Основанный на предположительных аминокислотах, кодируемых в нуклеотидной последовательности HCV1, небольшой домен во внешнем N-конце HCV полибелка, по-видимому, как по размеру, так и по высокому содержанию щелочных оснований подобен белку нуклеокапсида (C), находящемуся на N-конце флавивирусных полибелков. Неструктурные белки 2, 3, 4 и 5 (NS2-5) HCV и вируса желтой лихорадки (YFV), по-видимому, имеют противоположные части сходного размера и гидрофильности, хотя имеется дивергенция в аминокислотных последовательностях. Однако район HCV, который мог бы соответствовать районам YFV полибелка, которые содержат M, E и NS1 белки, не отличаются по последовательности, но, по-видимому, также совершенно отличны по размеру и гидрофильности. Таким образом, в то время как определенные домены генома HCV можно назвать как, например, NS1 или NS2, следует иметь в виду, что эти указания спекулятивные, между HCV семейством и флавивирусами могут быть значительные различия, которые еще придется оценить.
Ожидают, что различные штаммы, изоляты и субтипы HCV содержат различия в аминокислотах и нуклеиновых кислотах по сравнению с HCV1. Ожидают, что многие изоляты дадут большую гомологию (т.е. более чем около 40%) общей аминокислотной последовательности по сравнению с HCV1.
Однако может также оказаться, что есть другие менее гомологичные HCV изоляты. Их можно определить как HCV в соответствии с различными критериями, такими как, например, OPC с приблизительно 9000 нуклеотидов до приблизительно 12000 нуклеотидов, кодирующая полибелок с подобными HCV1 гидрофобным и/или антигенным свойствами, и присутствие построчно пептидных последовательностей, которые консервативны с HCV 1. В дополнение, считают, что геном позитивная цепь РНК.
Все HCV изоляты кодируют по крайней мере один эпитон, который иммунологически идентичен (т. е. иммунологически перекрестно реагирует) с эпитопом, кодируемым в HCV кДНК, описанной здесь. Предпочтительно этот эпитон включен в аминокислотную последовательность, здесь описанную, и является уникальным для HCV при сравнении с предварительно известными патогенами. Уникальность этого эпитона может определяться его иммунологической реактивностью и антиHCV-антителами и отсутствием иммунологической реакционной способности с антителами к известным патогенам.
HCV штаммы и изоляты являются эволюционно родственными. Следовательно, предполагают, что общая гомология геномов на нуклеотидном уровне возможно около 40% или выше, возможно будет около 50% или выше, возможно около 60% или выше и даже более возможно, что около 80% и выше, и в дополнение, что могут быть соответствующие прилежащие последовательности по крайней мере около 13 нуклеотидов. Следует отметить, как показано ниже, в HCV геноме есть вариабельные и гипервариабельные районы, следовательно, предполагают, что гомология этих районов значительно меньше, чем в целом по геному. Соответствие между предположительной геномной последовательностью цепи HCV и, например, кДНК последовательностью CDC/HCV1 можно определить с помощью методов, известных в науке. Например, их можно определить путем прямого сравнения информации о последовательности полинуклеотида предположительно HCV и HCV кДНК последовательностей, описанных здесь. Их можно также определить гибридизацией полинуклеотидов при условиях, при которых формируются стабильные дуплексы между гомологичными районами (например, те, что можно было бы использовать перед расщеплением S1), после чего следует расщепление нуклеазой, специфичной к одноцепочечной ДНК, затем следует определение величины переваренных фрагментов.
Вследствие эволюционной связи штаммов и изолятов HCV, предположительные HCV штаммы или изоляты идентичны по их гомологии на полипептидном уровне. В основном, предполагают, что HCV штаммы или изоляты гомологичны по крайней мере на 40%, гомологичны более 50%, возможно более чем около 70% гомологичны и даже более вероятно, что на приблизительно 90% гомологичны на полипептидном уровне. Технологии определения гомологии аминокислотной последовательности известны в науке. Например, аминокислотную последовательность можно определять непосредственно и сравнивать с последовательностями, приведенными здесь. Аналогично можно определить нуклеотидную последовательность геномного материала предположительно HCV (обычно через кДНК интермедиат), предположительную аминокислотную последовательность, кодируемую ей, и соответствующие районные сравнить.
Как здесь употребляется, полинуклеотид, "происходящий от" указанной последовательности, относится к полинуклеотидной последовательности, которая содержит в себе по крайней мере около 6 нуклеотидов, предпочтительно по крайней мере около 8 нуклеотидов, более предпочтительно по крайней мере около 10-12 нуклеотидов, и даже предпочтительно по крайней мере около 15-20 нуклеотидов, соответствующих району указанной нуклеотидной последовательности. "Соответствующий" подразумевает гомологичный или комплементарный обозначенной последовательности. Предпочтительно, чтобы последовательность района, из которого происходит полинуклеотид, являлась гомологичной или комплементарной уникальной в геноме HCV последовательности. Более предпочтительно, чтобы родственная последовательность была гомологичной или комплементарной уникальной последовательности во всех или огромном большинстве HCV изолятов. Уникальна последовательность в геноме HCV или нет, можно определить методами, известными тем, кто квалифицирован в этой области. Например, эту последовательность можно сравнить с последовательностями в банке данных, например в банке генов, чтобы определить, присутствует ли она в неинфицированных хозяевах или других организмах. Последовательность можно сравнить также с известными последовательностями других вирусных агентов, включая те, что, как известно, вызывают гепатиты, например HAV, HBV и HDV, и являются членами семейства Flaviviridae. Соответствие или несоответствие родственной последовательности другим последовательностям можно также выяснить гибридизацией при подходящих условиях жесткости. Методы гибридизации для определения комплементарности последовательностей нуклеиновых кислот в науке и обсуждаются здесь. См. также, например, Maniatis и др. (1982). В дополнение, неспаренные нуклеотиды в полинуклеотидном дуплексе, образованном при гибридизации, можно определить известными методами, включая, например, переваривание с нуклеазой, такой как S1, которая специфически расщепляет одноцепочечные области в полинуклеотидном дуплексе. Районы, из которых могут "происходить" типичные последовательности ДНК, включают (но не ограничиваются), например, районы, кодирующие специфические эпитоны, так же как нетранкскрибируемые и/или нетранслирумые районы.
Родственный полинуклеотид не обязательно физически происходит от показанной нуклеотидной последовательности, а может быть образован любым образом, включая, например, химический синтез, или репликацию ДНК, или обратную транскрипцию, или просто транскрипцию. В дополнение, комбинации районов, соответствующих районам указанной последовательности, можно модифицировать с помощью способов, известных в науке, чтобы согласовать их в свете предполагаемого использования.
Под термином "рекомбинантный полинуклеотид", как он здесь использован, подразумевается полинуклеотид геномный, кДНК, полисинтетический, синтетический, который по способу его происхождения или манипуляций с ним: 1) не имеет сходства с полным или частью полинуклеотида, с которым он ассоциируется в природе, 2) связан с другим полинуклеотидом, чем тот, с которым ассоциируется в природе, 3) не встречается в природе.
Термин "полинуклеотид", как он здесь использован, относится к полимерной форме нуклеотидов любой длины, либо рибонуклеотидов, либо дезоксирибонуклеотидов. Этот термин относится только к первичной структуре молекулы. Таким образом, этот термин включает одну- и двухцепочечные ДНК и РНК. Он также включает известные типы модификаций, например мечение, известное в науке, метилирование, "кэпирование", замены одного или более природных нуклеотидов на аналоги, межнуклеотидные модификации, такие как, например, модификации с неизмененными связями (например, метилфосфанаты, фосфотриэфиры, фосфоангидриды, карбаматы и т.д.) и с измененными связями (например, фосфоротиаты, фосфородитиаты и т. д.), содержащие "подвешенные" к ним части, например белки (включая например, нуклеазы, токсины, антитела, сигнальные пептиды, поли-L-лизин и т.д.) с интеркаляторами (например, акридин, псорален и т.д.), содержащие хелаты (например, металлы, радиоактивные металлы, бор, окисленные металлы и т. д. ), содержащие алкиляторы, с модифицированными связями (например, альфа аномерные нуклеиновые кислоты и т.д.), так же как немодифицированные формы этого полинуклеотида.
Как использовано здесь, "смысловая цепь" нуклеиновой кислоты содержит последовательность, которая имеет последовательность, гомологичную мРНК. "Антисмысловая цепь" содержит последовательность, комплементарную "смысловой цепи".
Как использовано здесь, "геном с позитивной цепью" вируса является геномом, состоящим из РНК либо ДНК, одноцепочечным, который кодирует вирусные полипептиды. Пример позитивных РНК вирусов включают тогавирусы, коронавирусы, ретровирусы, пикорнавирусы и калицивирусы. Также включены флавивирусы, которые прежде классифицировали как тогавирусы. См. Fields и Knipe (1986).
Термин "праймер", как здесь использовано, относится к олигомеру, который способен работать как участок инициации синтеза полинуклеотидной цепи, при инкубации в подходящих условиях. Праймер комплементарен или почти комплементарен району полинуклеотидной цепи, которую нужно скопировать. Таким образом, при условиях, ведущих к гибридизации, праймер отжигается с комплементарным районом цепи аналита. При добавлении подходящих реагентов (например, полимеразы, нуклеотид трифосфатов и подобных) праймер наращивается полимеризующими агентами, образуя копию цепи аналита. Праймер может быть одноцепочечным или альтернативно может быть частично или полностью двухцепочечным.
Термины "полинуклеотид аналита" и "цепь аналита" относятся к молекуле одно- или двухцепочечной нуклеиновой кислоты, которая, как подозревают, содержит последовательность мишени и которая может присутствовать в биологическом образце.
Как здесь использовано, термин "олигомер" относится к праймерам и пробам. Термин олигомер не имеет дополнительного значения длины молекулы. Однако обычно олигомеры не длиннее 1000 нуклеотидов, более типично не длиннее 500 нуклеотидов и даже более типично не длиннее чем 250 нуклеотидов, они могут быть не длиннее 100 нуклеотидов, и не длиннее чем 75 нуклеотидов, и также могут быть не более 50 нуклеотидов длины.
Как здесь использовано, термин "проба" относится к структуре, включающей полинуклеотид, который формирует гибридную структуру с последовательностью мишени, обусловленной комплементарностью по крайней мере одной последовательности в пробе с последовательностью в районе мишени. Полинуклеотидные районы проб могут состоять из ДНК, и/или РНК, и/или синтетических нуклеотидных аналогов. Со словом "проба" употребляются сочетания "захваченные пробы" и "меченые пробы". Предпочтительно, чтобы проба не содержала последовательность, комплементарную последовательности(тям), используемым в качестве праймеров для реакции наращивания цепи (PCR).
Как здесь использовано, термин "район мишени" относится к последовательности, с которыми проба или праймер формируют при требуемых условиях стабильный гибрид.
Термин "захватывающая проба", как здесь использовано, относится к полинуклеотиду, состоящему из одноцепочечного полинуклеотида, отожженного с связывающимся партнером. Одноцепочечный полинуклеотид включает полинуклеотидную последовательность для мишени, которая комплементарна последовательности мишени в районе мишени, чтобы быть детектированной в полинуклеотиде аналита. Этот комплементарный район достаточной длины и комплементарен последовательности мишени, чтобы позволить сформировать достаточно стабильный дуплекс, чтобы иммобилизовать полинуклеотид аналита на поверхности носителя (через связывающихся партнеров). Связывающийся партнер может быть связан с поверхностью твердого носителя или может быть связан непрямым способом через другие структуры или связывающихся партнеров с твердым носителем.
Термин "полинуклеотидная последовательность для мишени", как здесь использовано, относится к полинуклеотидной последовательности, которая включает нуклеотиды, комплементарные нуклеотидной последовательности мишени, эта последовательность достаточной длины и комплементарности с последовательностью мишени для образования дуплекса достаточной стабильности для намеченных целей.
Термин "связывающийся партнер", как здесь использовано, относится к молекуле, способной связывать молекулу лиганда с высокой специфичностью, как например антиген и специфичное ему антитело. В основном специфически связывающиеся партнеры должны связываться с достаточным сродством для иммобилизации дуплекса копия аналита/ комплементарная цепь (в случае захватывающих проб) при выбранных условиях. Специфически связывающиеся партнеры известны в науке и включают, например, биотин и авиадин или стрептавидин, IgG и белок A, многочисленные известные пары рецептор - лиганд и комплементарные полинуклеотидные цепи. В случае комплементарных полинуклеотидных связывающихся партнеров они обычно по крайне мере около 15 оснований в длину и могут быть по крайней мере 40 оснований длины, и, кроме того, они имеют F + C состав по крайней мере около 40% и максимально около 60%. Полинуклеотиды могут быть представлены ДНК, РНК или синтетическими нуклеотидными аналогами.
Термин "связанный", как здесь использовано, относится к способам присоединения посредством ковалентных связей или путем сильных нековалентных взаимодействий (например, гидрофобные взаимодействия водородные связи и т.д. ). Ковалентные связи могут быть, например, из сложных эфиров, простых эфиров, фосфоэфирными, амидными, пептидными, имидными, связи углерод-сера, связи углерод-фосфор и подобные.
Термин "носитель" относится к любой твердой или полутвердой поверхности, на которой можно заякорить требуемый связывающийся партнер. Подходящие подложки включают стекло, пластик, металл, полимерные гели и подобные и могут принимать форму гранул, лунок, мембран и подобных.
Термин "меченый", как здесь использовано, относится к любому атому или части, которые можно использовать для обеспечения детектируемого (предпочтительно количественного) сигнала и которые можно включить в полинуклеотид или полипептид.
Как здесь использовано, термин "меченая проба" относится к олигомеру, который включает полинуклеотидную последовательность для мишени, комплементарен последовательности мишени, которую нужно детектировать в полинуклеотиде аналита. Этот комплементарный район достаточной длины и комплементарен последовательности мишени, чтобы позволить образовать дуплекс, состоящий из "меченой пробы" и "последовательности мишени", который детектируется с помощью метки. Олигомер спаривается с меткой либо непосредственно, либо непрямым способом через набор молекул лигандов с высокой специфичностью друг к другу. Наборы молекул лигандов с высокой специфичностью описаны выше и также включают мультимеры.
Термин "мультимер", как здесь описано, относится к линейным или разветвленным полимерам одинаковой повторяющейся одноцепочечной полинуклеотидной единицы или разных одноцепочечных полинуклеотидных единиц. По крайней мере одна из единиц имеет последовательность, длину и состав, которые позволяют специфично гибридизоваться ей с первой же одноцепочечной нуклеотидной последовательностью, представляющей интерес, обычно аналитом или олигомером (например, меченой пробой), связанным с аналитом. Для того чтобы достичь такой специфичности и стабильности, эта единица обычно по крайней мере 15 нуклеотидов длины, типично не более около 50 нуклеотидов длины и предпочтительно около 30 нуклеотидов длиной, более того, F + Ц состав нормально по крайней мере около 40% и максимально около 60%. В дополнение к этой единице(ам) мультимер включает множество единиц, которые способны специфично и стабильно гибридизоваться со вторым одноцепочечным нуклеотидом, представляющим интерес, обычно меченым полинуклеотидом или еще одним мультимером. Эти единицы в основном приблизительно одинакового размера и состава с мультимерами, описанными выше. Когда мультимер предназначен для гибридизации с еще одним мультимером, первая и вторая олигонуклеотидные единицы гетерогенные (разные) и не гибридизуются друг с другом при выбранных условиях анализа. Таким образом, мультимеры могут быть мечеными пробами или лигандами, которые связывают метку с пробой.
Как здесь использовали, термин "вирусная РНК", которая включает HCV РНК, относится к РНК вирусного генома, его фрагментов, его транскриптов и происходящим из него мутантным последовательностям.
Как здесь использовано, "биологический образец" относится к образцу ткани или жидкости, изолированных из индивидуума, включающих (но не ограничивающихся), например, плазму, сыворотку, спиномозговую жидкость, лимфу, наружные покровы кожи, дыхательного, кишечного и мочеполового трактов, слезы, слюну, молоко, клетки крови, опухоли, органы и также образцы клеточной культуры in vitro (включающие, но не ограничивающиеся определенной средой, полученной при росте клеток в клеточной культуральной среде, предположительно инфицированные вирусом клетки, рекомбинантные клетки и клеточные компоненты).
Описание изобретения
Практика настоящего изобретения использует, если не оговорено особо, общепринятые технологии химии, молекулярной биологии, микробиологии, рекомбинантной ДНК и иммунологии, которые доступны квалифицированным в науке. Такие методы полностью даны в литературе. См., например, Maniatis, Fitsch и Sambrook, Molecular cloning; A Laboratory manual (1982); DNA cloning, Volumes I и II (D. N. Glover ed. 1985); oligonucleotide synthesis (M.I.Gaid ed., 1984); Nucleic acid hybridization (B.D.Hames S.I.Higgins eds. 1984); серии Methods in enzymology Academic press. Inc. ), особенно Vol 154 и Vol 155 (Wu and Grossman, and Wu, eds., соответственно). Все патенты, патентные заявления и публикации, о которых здесь упоминалось как выше, так и ниже, внесены здесь в ссылки.
Используемые материалы и процессы настоящего изобретения делают возможным обнаружение HSV как этиологического агента BB-NANBV и обеспечение семейством нуклеотидных последовательностей, выделенных из кДНК библиотек, которые содержат HCV кДНК последовательности. Эти кДНК библиотеки получали на основании последовательностей нуклеиновых кислот, присутствующих в плазме HCV-инфицированной шимпанзе. Конструирование одной из этих библиотек библиотеки "с" (АТСС N 40394) описано в БРО publication N 318216.
Используя вышеописанные HCV кДНК последовательности, можно сконструировать олигомеры, которые используют как реагенты для детекции вирусных полинуклеотидов в биологических образцах. Например, возможно синтезировать ДНК олигомеры длиной около 8-10 нуклеотидов или больше, которые используют как гибридизационные пробы для обнаружения присутствия HCV РНК, например, в крови доноров, фракциях крови, сыворотке индивидуумов, подозрительных на носительство вируса, или системах культуры клеток, в которых реплицируется вирус. Кроме того, новые олигомеры, описанные здесь, позволяют далее охарактеризовать HCV геном.
Полинуклеотидные пробы и праймеры, происходящие из этих последовательностей, можно использовать для амплификации последовательностей, присутствующих в кДНК библиотеках, и/или скринировать кДНК библиотеки для дополнительных перекрывающихся последовательностей кДНК, которые, в свою очередь, можно использовать для получения еще перекрывающихся последовательностей. Как указано ниже и в EPO publication N 318216, геном HCV, как кажется, РНК, включающая главным образом большую открытую рамку считывания (OPC), которая кодирует большой полибелок.
В дополнение к вышесказанному информация, представленная ниже, позволяет идентифицировать дополнительные HCV штаммы и изоляты. Выделение и характеристика дополнительных HCV штаммов или изолятов может быть выполнена используя техники, известные квалифицированным в этой области, например путем выделения нуклеиновых кислот из компонентов тела, которые содержат вирусные частицы и/или вирусную РНК, создания кДНК и библиотек, используя олигомеры, описанные ниже, для скрининга библиотек на клоны, содержащие HCV кДНК последовательности, описанные здесь, и сравнения HCV кДНК из новых изолятов с кДНК, описанными в EPO publication N 318216 и ниже. Легко идентифицируются штаммы и изоляты, которые подходят по параметрам к HCV, как описано в разделе определения. Другие методы идентификации штаммов очевидны для тех, кто квалифицирован в этой области, на основании информации, приведенной здесь.
Выделение HCV кДНК последовательностей
Олигомеры данного изобретения содержат районы, которые формируют гибридные структуры дуплексов с последовательностями для мишени в HCV полинуклеотидах. HCV полинуклеотидные гибридизирующиеся районы олигомеров можно установить из HCV кДНК последовательности(ей), приведенных здесь и описанных в EPO publication N 318216. Состав HCV кДНК из HCV1, прототипа HCV, показан на фиг. 18. Составная последовательность основана на информации о последовательности, берущей начало от целого ряда HCV кДНК клонов, которые были выделены из целого ряда HCV кДНК библиотек, включая "с" библиотеку, сделанную в лямбда-gt11 (АТСС N 40394) и из человеческой сыворотки. HCV кДНК клоны выделены с помощью методов, описанных EPO publication N 318216. Кратко, большинство клонов, которые были выделены, содержали последовательности из кДНК "с" библиотеки, которая была сконструирована используя набор сывороток из шимпанзе с хронической HCV инфекцией и содержащая высокий титр вируса, т.е. по меньшей мере 106 инфекционных доз шимпанзе/мл (CID/ml). Пул сывороток использовали для выделения вирусных частиц, нуклеиновые кислоты, выделенные из этих частиц, использовали как матрицу в конструировании кДНК библиотек вирусного генома. Первоначальный клон, 5-1-1, получен путем скрининга "с" библиотеки с сыворотками из инфицированных индивидуумов (с библиотеками). После выделения первого клона остаток последовательности был получен при скрининге с синтетическими полинуклеотидными пробами, последовательности которых происходили из 5'-района и 3'-района известной HCV кДНК последовательности/ей.
Описание методов восстановления кДНК последовательностей представляет главным образом исторический интерес. Результирующие последовательности (и их комплементы) приведены здесь, и последовательности или любая их часть могут быть приготовлены используя синтетические методы или путем комбинации синтетических методов с восстановлением частичных последовательностей, используя методы, подобные описанным в EPO publication N 318216.
Олигомерные пробы и праймеры
Используя как базис HCV геном (как иллюстрировано на фиг. 19) и/или предпочтительные консервативные районы HCV генома, можно приготовить олигомеры приблизительно 8 нуклеотидов или более длиной, которые гибридизуются с позитивной цепью(ями) РНК HCV или ее комплемента, так же как и с HCV кДНК. Эти олигомеры могут служить в качестве проб для детекции (включая выделение и/или мечение) полинуклеотидов, которые содержат HCV нуклеотидные последовательности, и/или как праймеры для транскрипции и/или репликации последовательностей HCV мишени. Олигомеры содержат полинуклеотидную последовательность для мишени, которая включает нуклеотиды, комплементарные нуклеотидной последовательности HCV мишени, а также последовательность достаточной длины и комплементарной HCV последовательности для формирования дуплекса, который достаточно стабилен для намеченных целей. Например, если целью является выделение через иммобилизацию аналита, содержащего HCV последовательность мишени, олигомеры будут содержать полинуклеотидный район, который имеет достаточную длину и комплементарность с HCV последовательностью мишени, чтобы обеспечить достаточную стабильность дуплекса для иммобилизации аналита на твердой поверхности через его связывание с олигомерами при условиях выделения. Например, также, если олигомеры служат в качестве праймеров для транскрипции и/или репликации HCV последовательностей мишени в полинуклеотиде аналита, олигомеры будут содержать полинуклеотидный район достаточной длины и комплементарности к HCV последовательности мишени, чтобы позволить полимеризующему агенту продолжать репликацию с праймеров, которые формируют стабильный комплекс с последовательностью мишени в условиях гибридизации. Например, также если олигомеры можно использовать в качестве меченых проб или связывать с мультимерами, полинуклеотидный район для связывания мишени должен быть достаточной длины и комплементарности для формирования стабильных гибридных дуплексных структур с мечеными пробами и/или мультимерами, чтобы позволить детектировать дуплекс. Олигомеры могут содержать как минимум около 4 прилежащих нуклеотидов, комплементарных HCV последовательности мишени, обычно олигомеры содержат минимум около 8 соседних нуклеотидов, комплементарных HCV последовательности мишени, и предпочтительно содержат минимум около 14 нуклеотидов, которые комплементарны HCV последовательности мишени.
Подходящая HCV нуклеотидная последовательность для мишени может включать нуклеотиды, комплементарные нуклеотидам, выбранным из HCV кДНК нуклеотидов (см. в конце описания), которые показаны на фиг. 18 (nnx-nny обозначают с приблизительно какого номера нуклеотида x по какой номер нуклеотида y).
Олигомер, однако, не обязательно состоит только из последовательности, комплементарной HCV последовательности мишени. Он может содержать дополнительно нуклеотидные последовательности или другие части, подходящие для тех целей, для которых используются олигомеры. Например, если олигомеры используются в качестве праймеров для амплификации HCV последовательностей через PCR, они могут содержать последовательности, формирующие в состоянии дуплекса сайты ферментов рестрикции, которые облегчают клонирование амплифицированных последовательностей. Например, также если олигомеры должны использоваться в качестве "захватывающих проб" в гибридизационных анализах (описанных ниже), они будут содержать в дополнение связывающий партнер, который связывается с олигомером, содержащим нуклеотидную последовательность, комплементарную HCV последовательности мишени. Другие типы мотивов и последовательностей, используемые в олигомерах или спаривающиеся с олигомерами, - последовательности, известные в науке и используемые в науке и используемые для различных целей, включая мечение нуклеотидных проб.
Приготовление олигомеров осуществляется посредством методов, известных в науке, например методами, которые включают эксцизию, транскрипцию или химический синтез. Последовательности мишени и/или районов генома, которые выбраны из олигомеров, которым комплементарны полинуклеотиды для мишени, зависят от нашей цели. Например, если цель - скринировать биологические образцы на присутствие HCV (например, кровь), предпочтительные олигомеры будут использоваться как пробы и/или праймеры и гибридизоваться с консервативными районами генома HCV. Некоторые из консервативных районов генома HCV, с которыми могут связываться олигомеры, описаны здесь, например районы, включающие нуклеотиды под номерами с 5'-терминального по приблизительно 200, или с 4000 по приблизительно 5000, или с 8000 по 9040, как показано на фиг. 18, или предпочтительно нуклеотиды - 318 по 174, 4056 по 4448 и 4378 по 4902. Другие районы генома, которые консервативны, легко устанавливаются при сравнении нуклеотидных последовательностей различных изолятов HCV, включая прототип HCV, HCV1.
Методы для проведения сравнений генотипов для определения консервативных и неконсервативных районов известны в науке, и примеры методов приведены здесь.
В основе гибридизационного анализа нуклеиновых кислот лежит гибридизация одноцепочечной нуклеиновой кислоты аналита (либо ДНК, либо РНК) с нуклеиновой кислотой пробы и полученные дуплексы детектируются. Пробы для HCV полинуклеотидов (естественные или производные) имеют длину, которая позволяет детектировать уникальные вирусные последовательности при гибридизации. В то время как "рабочая" длина может быть 6-8 нуклеотидов, последовательности в 10-12 нуклеотидов предпочтительны, и около 20 нуклеотидов и более, по-видимому, оптимальны. Лучше, чтобы эти последовательности происходили из районов с отсутствием гетерогенности. Эти пробы можно сделать, используя рутинные методы, включая автоматический метод синтеза олигонуклеотидов. Среди используемых проб, например, пробы, полученные из вновь выделенных клонов, приведенные здесь так же, как различные олигомеры, используемые как пробы с кДНК библиотеками, приведены ниже. Удовлетворительным будет комплемент к любой уникальной части HCV генома. Для использования в качестве проб желательна полная комплементарность, хотя это может быть необязательно, когда увеличена длина фрагмента.
Для использования таких проб как агентов для детекции присутствия HCV полинуклеотидов (например, при скрининге контаминированной крови) анализируемый биологический образец, такой как кровь или сыворотка, можно обработать, по желанию, чтобы экстрагировать нуклеиновые кислоты, содержащиеся в нем. Полученную из образца нуклеиновую кислоту можно разогнать электрофорезом в геле или подвергнуть другим технологиям разделения по размеру, с другой стороны, образец нуклеиновой кислоты можно перенести на блот, сделав доттинг, без разделения по размеру. Для формирования гибридных дуплексов с последовательностью для мишени пробы район для мишени нуклеиновой кислоты аналита должен быть в одноцепочечной форме. Там, где последовательность естественно присутствует в одноцепочечной форме, денатурация не требуется. Однако там, где последовательность присутствует в двухцепочечной форме, последовательность денатурируют. Денатурацию можно проводить различными методиками, известными в науке. Следом за денатурацией нуклеиновую кислоту аналита и пробу инкубируют при условиях, которые способствуют образованию стабильного гибрида последовательности мишени в пробе и предположительно последовательности для мишени в аналите, и полученные дуплексы, содержащие пробу(ы), детектируют.
Детекцию результирующего дуплекса или любого другого обычно осуществляют путем использования меченых проб, альтернативно проба может быть немеченой, но детектироваться специфическим связыванием с лигандом, который помечен либо прямо, либо косвенно. Подходящие метки и методы мечения проб и лиганды известны в науке и включают, например, радиоактивные метки, которые можно включить известными методами (например, ник-трансляцией или киназой), биотин, флуоресцентные группы, хемилюминесцентные группы (например, диокситаны, особенно триггердиокситаны), ферменты, антитела и другие.
Район проб, который используется для связывания аналита, может быть полностью комплементарен HCV геному. Следовательно, обычно желательны очень жесткие условия (гибридизации) для того, чтобы избежать ошибочных позитивных сигналов. Однако жесткие условия следует использовать, только если пробы комплементарны районам вирусного генома с отсутствием гетерогенности. Жесткость гибридизации определяют целый ряд факторов во время гибридизации и во время отмывки, включая температуру, ионную силу, продолжительность, концентрацию формамида. Эти факторы отмечены, например, в Maniatis, T (1982).
Отклонения от этой основной схемы, принятой в науке, включая те, что облегчают отделение дуплексов, которые надо детектировать от других материалов, и/или те, что усиливают сигнал от меченого мотива, также можно использовать. Целый ряд этих модификаций обсуждается, например, y: Matthews и Kricka (1988), Analytical Biochemistry 169:1, Landegreu и др. (1988), Science 242:229, и Mittlin (1989), Clinical chem, 35: 1819. Эти и нижеследующие публикации, описывающие обширность вариаций анализа, вынесены здесь в ссылки. Подходящие для детекции HCV в этих анализах пробы включают последовательности, гибридизующиеся с полинуклеотидными последовательностями HCV мишени для формирования дуплексов с цепью аналита, где дуплексы обладают достаточной стабильностью для детекции в специфической системе анализа.
Подходящая модификация описана, например, в US patent N 4868105, вышедшего 9 сентября 1989, и EPO publication N 295807 (опубликована 16 июня 1987). Эти публикации описывают метод гибризации нуклеиновых кислот в жидкой фазе, в котором нуклеиновую кислоту аналита гибридизуют с набором меченых проб с набором захватывающих проб. Комплекс проба - аналит связывается при гибридизации с захватывающей пробой на твердом носителе, которая комплементарна набору захватывающих проб. Это позволяет удалять нуклеиновую кислоту аналита из раствора как твердофазный комплекс. Имея аналит в форме твердофазного комплекса, это облегчает последующие стадии его отделения в анализе. Набор меченых проб комплементарен меченой пробе, которая связывается посредством гибридизации с комплексом твердая фаза/аналит.
В большинстве случаев ожидают, что последовательности генома будут присутствовать в сыворотке инфицированных индивидуумов на относительно низких уровнях, т. е. приблизительно 102 - 103 инфекционных доз шимпанзе (CID) на мл. Такой уровень, возможно, потребует использования амплификационных методик в гибридизационных анализах. Такие технологии известны в науке. Например, система Enzo Biochemical Corporation "Bio-Bridge" использует трансферазу терминальных дезоксинуклеотидов для добавления немодифицированных 3'-поли-дТ-хвостов к ДНК пробе. Поли дТ-концевая проба гибридизуется с нуклеотидной последовательностью мишени и затем с биотинмодифицированным поли-А. PCT publication 84/03520 и EP publication N 124221 описывают гибридизационный анализ ДНК, в котором: 1) аналит отжигается с одноцепочечной пробой ДНК, комплементарной меченому ферментом олигонуклеотиду, и 2) полученный дуплекс с хвостом гибридизуется с ферментмеченым олигонуклеотидом, EPA 204510 описывает гибридизационный анализ ДНК, в котором ДНК аналита контактирует с пробой, которая имеет хвост, такой как поли-дТ хвост, и цепь амплификатора, которая имеет последовательность, гибридизующуюся с хвостом пробы, такой как последовательность поли-А, и которая способна связывать множество меченых цепей. Тип гибридизационного анализа, который описан в EPO publication N 317 077 (опубликованном 24 мая 1989), который детектирует последовательности на уровне приблизительно 106/мл, использует мультимеры нуклеиновых кислот, которые связываются с одноцепочечной нуклеиновой кислотой аналита и которые также связываются со множеством одноцепочечных меченых олигонуклеотидов. Особенно предпочтительная технология, возможно, включает амплификацию последовательностей HCV мишени в сыворотке приблизительно в 10000 раз (т. е. приблизительно 106 последовательностей/мл) как часть гибридизационной системы. Амплификацию можно осуществить полимеразной цепной реакцией (PCR), описанной Saiki и др. (1986), Mullis, US patent N 4683195 и Mullis и др. , US patent N 4683202. Амплификация может предшествовать или предпочтительно следовать перед очисткой HCV последовательностью мишени. Например, амплификацию можно использовать вместе с методами анализа, описанными в US patent N 4868105 или если желательна даже дальнейшая амплификация, в совокупности с гибридизационной системой, описанной в EPO publication N 317077. Предпочтительные методы для обнаружения HCV последовательностей в полинуклеотидной цепи аналита основаны на методах определения при помощи гибридизации, описанных в US patent N 4868105 и EPO publication N 317077. Эти методы - гибридизационные анализы в водной фазе по типу "сэндвича", которые используют как захватывающие, так и меченые пробы, которые гибридизируются с последовательностями, мишени в нуклеиновой кислоте аналита. При использовании этих анализов для скрининга биологических образцов на HCV используемые пробы связываются с консервативными районами генома. Захватывающие и меченые пробы можно объединять при связывании их с последовательностью мишени. Альтернативно в предпочтительном способе захватывающая и меченая пробы имеются в наборах и пробы одного набора не мешают с пробами другого набора. И в последнем способе предпочтительно набор(ы) множественных захватывающих проб гибридизовать с наиболее консервативными районами генома, в то время как набор(ы) множества меченых проб можно гибридизовать с районами, которые демонстрируют небольшую дивергенцию. Например, используя прототип HCV1 кДНК последовательность, показанную на фиг. 18, можно было бы использовать пробы, которые гибридизуются с последовательностями в районах нуклеотидов с приблизительно -318 по 174 и/или нуклеотиды в районе около 4378 по около 4902, и/или нуклеотиды в районе с 4056 по 4448. Предпочтительные пробы гибридизуются с последовательностями в 5'-районе HCV генома, так как, как показано внизу, этот район, по-видимому, высококонсервативен. Таким образом, предпочтительные пробы могут гибридизоваться с либо позитивной цепью в консервативных районах, и/или ее комплементом в зависимости от цели, например, чтобы обнаружить вирусные геномные последовательности, либо чтобы детектировать HCV кДНК последовательности, полученные амплификацией, или чтобы детектировать репликативные интермедиаты к позитивной HCV РНК цепи.
Обнаружение РНК HCV и берущих начало с нее полинуклеотидов, используя метод HCV/c PCR
Особенно полезным методом для обнаружения PHK HCV или родственных HCV РНК полинуклеотидов является HCV/cPCR метод, который является предметом этого заявления и который использует технологию полимеразной цепной реакции (PCR), описанную Saiki и др. (1986), Mullis in US pat. N 4683195 и Mullis и др. in US patent N 4683202, HCV/cPCR метод использует праймеры и пробы, имеющиеся в приведенной здесь информации, касающейся природы HCV генома.
В большинстве случаев в технике PCR готовят короткие олигонуклеотидные праймеры, которые спариваются c противоположными концами желаемой последовательности. Последовательность между праймерами знать не нужно. Образец полинуклеотида экстрагируют и денатурируют, лучше нагреванием, и гибридизуют с олигонуклеотидными праймерами, присутствующими в молярном избытке. Полимеризация катализуется образец- и праймерзависимой полимеразой в присутствии дезоксинуклеотид трифосфатов и аналогов нуклеотидов (дНТФ). Это приводит к двум "длинным продуктам", которые содержат соответствующие праймеры в качестве тх 5-конца, ковалентно связанные с новосинтезированными комплементарными цепями первоначальной цепи. Реплицированную ДНК снова денатурируют, гибридизуют с олигонуклеотидными праймерами, восстанавливают условия гибридизации и начинают второй цикл репликации. Второй цикл обеспечивает две начальных цепи, два длинных продукта из цикла 1 и два "коротких продукта", реплицированных с длинных. Короткие продукты содержат последовательности (смысловые или антисмысловые), берущие начало от последовательности мишени, фланкированной на 5'- и 3'-концах последовательностями праймера. В каждом дополнительном цикле количество коротких продуктов реплицирующихся возрастает экспоненциально. Таким образом, этот процесс вызывает амплификацию специфической последовательности мишени.
В этом методе поставляется образец, подозрительный на содержание HCV РНК, или ее фрагмент. Образец обычно забирается у индивидуума, подозрительного на NANBH, однако другие источники образца включают, например, определенную среду или клетки из in vitro систем, в которых реплицируется вирус. Образец, однако, должен содержать последовательность(и) нуклеиновой кислоты мишени.
Этот образец затем помещают в условия, позволяющие превратить с помощью обратной транскрипции HCV РНК в HCV кДНК. Условия для обратной транскрипции РНК известны тем, кто квалифицирован в этой области, и описаны, например, в Maniatis и др. (1982) и в Methods in Enzymology. Один предпочтительный метод обратной транскрипции использует обратную транскриптазу из множества источников, включая рекомбинантные молекулы и выделенные, например, из ретровирусов, преимущественно из вируса миелобластоза птиц (AMV), и подходящие условия для транскрипции. HCV кДНК продукт обратной транскрипции является РНК: ДНК гибридом, который получается при первом раунде обратной транскрипции, ДНК:ДНК гибриды образуются при двух или более раундах транскрипции.
HCV кДНК, образующийся при обратной транскрипции, затем подвергается PCR для амплификации последовательности мишени. Для амплификации HCV кДНК денатурируют и разделенные цепи гибридизуют с праймерами, фланкирующими последовательность мишени.
Разделение цепей можно получить любым подходящим методом денатурации, включая физические, химические и энзиматические, известные тем, кто квалифицирован в этой области. Предпочтительный метод, который является физическим, включает нагревание нуклеиновой кислоты до тех пор, пока она полностью (99%) не денатурирует. Типичная тепловая денатурация включает разброс температур от около 80oC до около 105oC и время от 1 до 10 минут.
После гибридизации HCV кДНК с праймерами последовательности мишени HCV реплицируют посредством полимеризации, которая использует какой-либо олигонуклеотидный праймер, чтобы инициировать синтез репликативной цепи. Праймеры отбирают так, чтобы они были комплементарны последовательностям HCV генома. Олигомерные праймеры, комплементарные районам смысловой и антисмысловой цепей HCV кДНК, могут быть составлены (скопированы) с HCV кДНК последовательностей из составной последовательности кДНК, приведенной на фиг. 18.
Праймеры выбираются так, чтобы их положения относительно последовательности дуплекса были такими, чтобы наращиваемый продукт, синтезированный с одного праймера, при отделении его от матрицы (комплемента) служил в качестве матрицы для наращивания другого праймера, чтобы дать репликативную цепь определенной длины.
Праймер является предпочтительно одноцепочечным для максимальной эффективности амплификации, но может быть альтернативно и двухцепочечным. Если праймер двухцепочечный, его сначала нагревают для разделения его цепей перед использованием для получения продуктов наращивания цепи. Лучше если праймер является олигодезоксирибонуклеотидом. Праймер должен быть достаточно длинным для праймирования синтеза продуктов наращивания в присутствии агентов полимеризации. Точные длины праймеров будут зависеть от многих факторов, включая температуру и источник праймера и способ использования этого метода. Например, в зависимости от сложности последовательности мишени олигонуклеотидный праймер обычно содержит около 15-45 нуклеотидов, хотя может содержать больше или меньше нуклеотидов. Молекулы коротких праймеров в основном требуют более низких температур, чтобы эффективно образовывать стабильные гибридные комплексы с матрицей.
Используемые здесь праймеры отбирают таким образом, чтобы они были "в значительной мере" комплементарны различным цепям каждой специфической последовательности, которую надо амплифицировать. Следовательно, праймерам не надо отражать точную последовательность матрицы, но они должны быть достаточно комплементарными, чтобы селективно гибридизоваться с соответствующими им цепями. Например, некомплементарный нуклеотидный фрагмент можно присоединить к 5-концу праймера, причем остаток последовательности праймера комплементарен определенной цепи. С другой стороны, некомлементарные основания или более длинные последовательности можно вводить в праймер при условии, что праймер достаточно комплементарен одной из цепей для амплификации при гибридизации с ней и вследствие этого для формирования структуры дуплекса, которую можно наращивать посредством полимеризации. Некомплементарные нуклеотидные последовательности праймеров могут включать сайты ферментов рестрикции. Добавление сайта рестрикции к концу(ам) последовательности мишени было бы особенно полезным для клонирования последовательности мишени.
Имейте в виду, что "праймер", как здесь использовано, может относиться к более чем одному праймеру, особенно в случае, где имеется некоторая двусмысленность в информации, касающейся концевых последовательностей(и) района мишени, которую амплифицируют. Однако "праймер" включает коллекцию олигонуклеотидных праймеров, представляющих возможные вариации в данной последовательности или включает нуклеотиды, которые допускают типичное спаривание. Один из олигонуклеотидных праймеров этой коллекции гомологичен концу последовательности мишени. Специфический случай показан в примерах, где олигомерные наборы 44-меров и 45-меров использовали, чтобы инициировать амплификацию потенциально вариантного района HCV генома.
Ожидают, что найдется большое разнообразие штаммов или изолятов HCV с последовательностями, отличающимися от HCV1, штамма-прототипа. Следовательно, для того чтобы детектировать различные штаммы, лучше конструировать праймеры, которые гибридизуются с консервативными районами HCV генома. Консервативные районы можно определить при сравнении нуклеотидной и аминокислотной последовательностей нескольких HCV штаммов/изолятов. По-видимому, есть по меньшей мере три района в HCV геноме консервативных аминокислот, описанных выше, из которых могут происходить праймеры. Считается, что такие районы есть. Праймеры, описанные ниже, в примерах, берут начало из, как считают, консервативных районов HCV, основываясь на гомологии последовательностей с последовательностями флавивирусов.
Олигонуклеотидные праймеры можно приготовить любым из подходящих методов. Методы получения олигонуклеотидов специфической последовательности известны в науке и включают, например, клонирование и расщепление рестриктазой подходящих последовательностей и прямой химический синтез. Методы химического синтеза могут включать, например, фосфотриэфирный метод, описанный Narang и др. (1979), фосфодиэфирный метод, открытый Brown и др. (1979), диэтилфосфоамидатный метод, открытый Beaucage и др. (1981) и метод на твердом носителе из US patent N 4458066.
Праймеры могут быть мечены, по желанию, способами включения (метки), детектируемой спектроскопически, фотохимически, биохимически, иммунохимически или химически.
Матрицезависимое наращивание олигонуклеотидного праймера/ов катализируется полимеризующими агентами в присутствии адекватных количеств четырех дезоксирибонуклеотидтрифосфатов (дАТФ, дЦТФ и дГТФ, дТТФ) или их аналогов, в реакционной среде, которая состоит из подходящих солей, катионов металла и забуференной системы с определенной pH. Подходящими полимеризующими агентами являются ферменты, о которых известно, что они катализируют праймер- и матрицезависимый синтез ДНК. Известные ДНК-полимеразы включают, например, ДНК-полимеразу IE coli или ее фрагмент Кленова, Т4 ДНК-полимеразу и Tag ДНК-полимеразу. Условия реакции для катализа синтеза ДНК с этими ДНК-полимеразами известны в науке.
Продукты такого синтеза - молекулы дуплексов, состоящие из цепей матриц и цепей наращиваемого праймера, которые включают последовательность мишени. Эти продукты, в свою очередь, служат как матрицы для еще одного раунда репликации. Во втором раунде репликации наращиваемая праймером цепь первого цикла отжигается со своим комплементарным праймером, синтез дает "короткий" продукт, ограниченный как на 5'-, так и на 3'-конце последовательностями праймера или его комплементами. Повторяющиеся циклы денатурации, отжига праймера и элонгации приводят к экспоненциальному накоплению района мишени, заданной праймерами. Достаточное количество циклов проводят, чтобы достигнуть желаемого количества полинуклеотида, содержащего район мишени нуклеиновой кислоты. Желаемое количество может варьировать и определяется задачей, которой служит полинуклеотидный продукт.
Метод PCR можно проводить на целом ряде последовательностей матриц. Например, он может выполняться поэтапно, когда после каждой стадии добавляются новые реагенты, или способом, когда все реагенты добавлены одновременно, или частично поэтапной методикой, когда свежие реагенты добавляются после определенного количества стадий.
Предпочтительный метод реакции PCR осуществляется как автоматизированный процесс, использующий термостабильный фермент. В этом процессе реакционная смесь проводится последовательно через отсек денатурации, отсек отжига праймера и отсек реакции. Можно использовать машину, которая специально приспособлена для использования с термостабильным ферментом, которая дает смену температур без водяной регулируемой вручную системы, так как не надо добавлять фермент в каждом цикле. Такой тип машины - подходящий коммерческий продукт Perkin Elmer Cetus Corp.
После амплификации с помощью PCR полинуклеотиды мишени обнаруживаются гибридизацией с полинуклеотидной пробой, которая формирует стабильный гибрид с последовательностью мишени при жестких или до умеренно жестких условиях гибридизации и отмывки. Если ожидают, что пробы будут полностью комплементарны (т.е. около 99% или выше) последовательности мишени, используют жесткие условия. Если ожидают несколько замен, например, если ожидают вариантные штаммы, которые приведут к тому, что проба не будет полностью комплементарна, жесткость гибридизации может быть уменьшена. Однако выбирают такие условия, которые исключают неспецифическое/случайное связывание. Условия, которые влияют на гибридизацию и которые выбирают, чтобы исключить неспецифическое связывание, известны в науке и описаны, например, Maniatis и др. (1982). В целом более низкая солевая концентрация и высокая температура увеличивают жесткость связывания. Например, обычно считается, что жесткие условия инкубации - в растворах, которые содержат приблизительно 0,1 х SSC, 0,1% ДДС, при около 65oC температуре инкубации/отмывки, и умеренно жесткие условия - инкубация в растворах, которые содержат приблизительно 1-2х SSC, 0,1% ДДС и температура отмывки/инкубации около 50-65oC. Низкая жесткость условий - 2х SSC и около 30-50oC.
Пробы для HCV последовательностей мишени могут происходить от HCV кДНК последовательности, показанной на фиг. 18 или из новых изолятов HCV. HCV пробы могут быть любого подходящего размера, который охватывает район мишени, но включает праймеры и который дает специфическую гибридизацию с районом мишени. Если есть полная комплементарность, т.е. если штамм содержит последовательность, идентичную пробу, так как дуплекс будет относительно стабильный при даже жестких условиях, пробы могут быть короткие, т.е. в пределах около 10-30 пар нуклеотидов. Если ожидают некоторый процент замен с пробой, т. е. если сомневаются, что проба будет гибридизироваться с вариантным районом, проба может быть большей длины, так как, по-видимому, длина несколько смягчает эффект неспаренных замен. Как пример этого приводится в примерах, где описаны пробы, которые связываются с потенциальными вариантами HCV1. В этом случае праймеры, предназначенные для связывания с HCV кДНК, происходили из гипотетически консервативного района HCV генома, и один из районов, содержащих потенциально вариации (основываясь на модели фравивирусов), был районом мишени. Проба, используемая для детекции последовательностей мишени, содержала приблизительно 268 пар нуклеотидов.
Нуклеиновую кислоту пробы, имеющую последовательность, комплементарную последовательности мишени, можно синтезировать, используя подобные методики, описанные выше, для синтеза последовательностей праймеров. По желанию пробу можно пометить. Подходящие метки описаны выше.
В некоторых случаях может быть желательным определить размер PCR продукта, детектированного пробой. Это может быть особенно важно, если подозревают, что вариантные HCV штаммы содержат делеции внутри района мишени, или если желают подтвердить длину PCR продукта. В таких случаях лучше провести анализ размера продуктов, а также гибридизацию с пробой. Методы определения размера нуклеиновых кислот известны в науке и включают, например, электрофорез в геле, осаждение в градиентах и хроматографию.
Присутствие последовательности мишени в биологическом образце детектируют путем определения, образуется ли гибрид между HCV полинуклеотидной пробой и нуклеиновой кислотой, подверженной технологии PCR амплификации. Методы для детекции гибридов, образованных пробой и нуклеиновой кислотой, известны в науке. Например, общепринято, что немеченый образец можно перенести на твердый матрикс, с которым он связывается, и связывающийся образец подвергнуть гибридизации в условиях, способствовавших специфической гибридизации с меченой пробой, твердый матрикс затем исследуется на присутствие меченой пробы. Альтернативно если образец помечен, немеченую пробу связывают с матриксом и после экспозиции в подходящих условиях гибридизации матрикс исследуется на присутствие метки. Другие подходящие методы гибридизации описаны выше.
Определение вариантных HCV последовательностей, используя PCR
Для того чтобы идентифицировать HCV штаммы и тем самым обозначить пробы для этих вариантов, используют вышеописанный HCV/cPCR метод, чтобы амплифицировать вариантные районы генома, так что можно определить нуклеиновые последовательности этих вариантных районов мишени. Главным образом вариантные типы HCV, как можно было бы ожидать, встречаются в других географических районах, чем те, в которых преобладает HCV1 штамм, например Японии, Африке и т.д., или в различных видах позвоночных, которые также инфицированы вирусом. Вариантный HCV может также возникать во время пассажа в системах культуры тканей или быть результатом спонтанных или индуцированных мутаций.
Для того чтобы амплифицировать вариантный район мишени, конструируют праймеры, чтобы фланкировать подозрительный район, и предпочтительно комплементарные консервативным районам. Праймеры к двум районам HCV, которые, возможно, консервативны, на основании модели флавивирусов, описаны в примерах. Эти праймеры и пробы можно приготовить, используя информацию о последовательностях HCV1 штамма, приведенную на фиг. 18.
Анализ нуклеотидной последовательности района/ов мишени может быть прямым анализом амплифицированных продуктов PCR. Процесс непосредственного анализа последовательности амплифицированных продуктов PCR описан у Saiki и др. (1988).
Альтернативно, амплифицированную последовательность/и мишени можно клонировать перед анализом последовательности. Метод прямого клонирования и анализа последовательности энзиматически амплифицированных геномных фрагментов описан Scharf (1986). В этом методе используемые в технологии PCR праймеры модифицированы возле их 5'-концов для того, чтобы дать подходящие рестрикционные сайты для клонирования непосредственно в, например, М13 вектор для сиквенса. После амплификации продукты PCR расщепляют подходящими ферментами рестрикции. Рестрикционные фрагменты лигируют с М13 вектором и трансформируют, например, JM 103 хозяина, высевают на чашки и полученные клоны скринируют гибридизацией с меченой олигонуклеотидной пробой. Другие методы клонирования и анализа последовательности известны в науке.
Универсальные праймеры для флавивирусов и HCV
Изучение природы генома HCV, используя пробы, происходящие из HCV кДНК, так же, как и информация, содержащаяся в HCV кДНК, предлагает, что HCV является флавиподобным вирусом. Эти исследования описаны в EPO publication N 318216, имеющихся у настоящего уполномоченного, которые вынесены здесь во всей их полноте. Сравнение HCV кДНК последовательности, родственной HCV кДНК клонам, с известными последовательностями целого ряда флавивирусов показывает, что HCV содержит последовательности, гомологичные консервативным последовательностям флавивирусов. Эти консервативные последовательности могут позволить создать праймеры, которые могут быть универсальны в приложении для амплификации районов мишени флавивирусов и HCV. Эти последовательности - 16-меры и меньшие последовательности от 3'-концов праймеров, описанных в примерах. Затем выполняют идентификацию видов, используя пробу, специфичную к этим видам. Геномы целого ряда флавивирусов известны в науке и включают, например, вирус японского энцефалита (Sumiyoshi и др., 1987), вирус типа 2 лихорадки денге (Hahn и др. , 1988), вирус желтой лихорадки (Rice и др., 1985), вирус лихорадки денге типа 4 (Mackow, 1987), вирус Западного Нила (Castle и др. , 1986). Идентификация РНК HCV выполняется, используя пробу, специфичную для HCV, последовательность которой можно установить из HCV кДНК последовательностей, приведенных здесь.
Альтернативно, использование наборов пробы, предназначенных для объяснения вырожденности кодонов и, следовательно, содержания общих последовательностей флавивирусами и HCV, как определено сравнением аминокислотных последовательностей HCV с известными последовательностями флавивирусов, позволяет иметь основную систему детекции для этих вирусов.
Конструирование желаемых последовательностей ДНК
Синтетические олигонуклеотиды можно приготовить используя автоматический олигонуклеотидный синтезатор, как описано Warner (1984). Если желательно, синтетические цепи можно пометить 32P путем обработки полинуклеотид киназой в присутствии 32P-АТФ, используя стандартные условия реакции.
Последовательности ДНК, включая выделенные из кДНК библиотек, можно модифицировать известными технологиями, включая, например, сайтнаправленный мутагенез, как описано Zoller (1982). Вкратце, ДНК, которую надо модифицировать, упаковывают в фаг как одноцепочечную последовательность и превращают в двухцепочечную ДНК, используя ДНК-полимеразу и как праймер синтетический олигонуклеотид, комплементарный части модифицируемой ДНК, и имея желаемую модификацию включенной в его собственную последовательность. Полученной двухцепочечной ДНК трансформируют в поддерживающую фаг бактерию-хозяина. Культуры трансформированных бактерий, которые имеют репликации каждой цепи фага, высевают на агар для получения бляшек. Теоретически 50% новых бляшек содержит фаг, имеющий мутантную последовательность, и оставшиеся 50% сохраняет первоначальную последовательность. Реплики с этих чашек гибридизуют с меченой синтетической пробой при температурах и условиях, которые допускают гибридизацию с нужной цепью, но не с немодифицированной последовательностью. Идентифицированные гибридизацией последовательности обнаруживаются и клонируются.
Киты для скрининга HCV родственных полинуклеотидов
Олигомеры - пробы и/или праймеры для амплификации и/или скрининга образцов на HCV можно выпускать как киты (наборы реагентов). Киты для скрининга на HCV последовательности включают ДНК олигомерных проб. Киты для амплификации последовательностей HCV могут включать олигомерные праймеры, используемые для амплификации. Обычно киты содержат пробы или праймеры в предварительно измеренных или предварительно определенных количествах так же, как и другие подходящие распакованные реагенты и материалы, в отдельных подходящих контейнерах, необходимых для определенной гибридизации и/или описания(ий) амплификации. Например, кит может содержать стандарты, буферы, подложки, ферменты, субстраты, меченые пробы, связывающиеся партнеры и/или инструкции для проведения теста.
Примеры
Внизу описаны примеры настоящего изобретения, которые приводятся только для иллюстративных целей и не ограничивают масштабы настоящего изобретения.
Выделение и последовательности перекрывающихся кДНК клонов 13i, 26j, CA59a, CA84a, CA156e и CA167b
Клоны 13i, 26j, CA59a, CA84a, CA156e и CA167b были выделены из лямбда-gt11 библиотеки, которая содержит HCV кДНК (АТСС N 40394), приготовление которой описано в EPO publication N 318216 (опубликовано 31 мая 1989) и WO 89/04669 (опубликовано 1 июня 1989). Скрининг библиотеки проводился с пробами, описанными ниже, используя метод, описанный Huynh (1985). Частота появления с соответствующими пробами позитивных клонов была около 1 из 50000.
Выделение клона 13i выполнялось, используя синтетическую пробу, производную от последовательности клона 12f. Последовательность этой пробы была:
5' GAA CGT TGC GAT CTG GAA GAC AGG GAC AGG3'.
Выделение клона 26j выполнялось, используя пробу, производную из 5'-района клона К9-1. Последовательность пробы была:
5'TAT CAG TTA TGC CAA CGG AAG CGG CCC CGA 3'.
Процедуры выделения клона 12f и клона k9-1 (также обозначенным K9-1) описаны в EPO publication N 318216 и их последовательности приведены на фиг. 1 и 2 соответственно. HCV кДНК последовательности клонов 13i и 26j показаны на фиг. 4 и 5 соответственно. Также показаны аминокислоты, кодируемые ими так же, как перекрвание клона 13i с клоном 12f и перекрывание клона 26j с клоном 13i. Последовательности этих клонов подтвердили последовательность клона K9-1. Клон K9-1 был выделен из другой HCV кДНК библиотеки (см. EPO publication N 218316).
Клон CA59a выделене, используя пробу, основанную на последовательности 5'-района клона 26. Последовательность этой пробы была:
5' CTG GTT AGC AGG GCT TTT CTA TCA CCA CAA 3'.
Проба, производная от последовательности клона CA59a, использовалась для выделения клона CA84a. Последовательность пробы, использованной для этого выделения, была:
5' AAG GTC CTG GTA GTG CTG CTG CTA TTT GCC 3'.
Клон CA156e выделен, используя пробу, происходящую из последовательности клона CA84a. Последовательность пробы была:
5' ACT GGA CGA CGC AAG GTT GCA ATT GCT CTA 3'.
Клон CA167b выделен, используя пробу, производную от последовательности клона CA156e. Последовательность пробы была:
5' TTC GAC GTC ACA TCG ATC TGC TTG TCG GGA 3'.
Нуклеотидные последовательности HCV кДНК в клонах CA59a, CA84a, CA156e и CA167b показаны на фиг. 6, 7, 8 и 9 соответственно. Аминокилсоты, кодируемые ими так же, как перекрывание с последовательностями соответствующих клонов, также показаны на этих чертежах.
Создание "pi" HCV кДНК библиотеки
Библиотека HCV кДНК, "pi" библиотека, была сконструирована из плазмы инфицированной шимпанзе того же сорта, использованной для конструирования лямбда-gt11 HCV кДНК библиотеки (АТСС N 40394), описанной в EPO publication N 318216 и иcпользующей преимущественно те же технологии. Однако при создании pi библиотеки использовали метод наращивания праймера, в котором праймер для обратной транскрипции основывался на последовательности клона CA59a. Последовательность праймера была:
5' GGT GAC GTG GGT TTC 3'.
Выделение и последовательность клона pi 14a
Скрининг "pi" HCV кДНК библиотеки описан выше с пробой, использованной для выделения клона CA167b (см. выше), давшим клон pi 14a. Этот клон содержит около 800 пар оснований кДНК, которые перекрывают клоны CA167b, CA156e, CA84a и CA59a, которые выделили из лямбда-gt11 HCV кДНК библиотеки (АТСС N 40394). Дополнительно pi 14a также содержит около 250 пар оснований ДНК, расположенной выше HCV кДНК из клона CA167b.
Выделение и последовательность клонов CA216a, CA290a и ag30a
На основании последовательности клона CA167b была сделана синтетическая проба, имеющая следующую последовательность:
5' GGC TTT ACC ACG TCA CCA ATG ATT GCC CTA 3'.
Вышеприведенную пробу использовали, чтобы скринировать HCV последовательности, что дало клон CA216a, показанные на фиг. 10.
Еще одну пробу сделали на основании последовательности клона CA216a, имеющего следующую последовательность:
5' TTT GGG TAA GGT CAT CGA TAC CCT TAC GTG 3'.
Скрининг лямбда-gt11 библиотеки (АТСС N 40394) с этой пробой дало клон CA290a, HCV последовательности которого представляются на фиг. 11.
В параллельном подходе делали праймернаращиваемую кДНК библиотеку, используя нуклеиновые кислоты, экстрагированные из той же инфицированной плазмы, использованной в оригинальной лямбда-gt11 кДНК библиотеке, описанной выше. Использованный праймер был основан на последовательности клонов CA216a и CA290a:
5' GAA GCC GCA CGT AAG 3'.
кДНК библиотеку сделали, используя методы, подобные описанным ранее для библиотек, использованных при выделении клонов pi14a и K9-1. Проба, использованная для скрининга этой библиотеки, была основана на последовательности клона CA290a:
5' CCG GCG TAG GTC GCG CAA TTT GGG TAA 3'.
Клон ag30a изолирован из новой библиотеки с вышеприведенной пробой и содержал около 670 пар оснований последовательности HCV. См. фиг. 12. Часть этой последовательности перекрывается с HCV последовательностью клонов CA216a и CA290a. Около 300 пар оснований последовательности ag30a, однако, лежит выше последовательности клона CA290a. Неперекрывающаяся последовательность показывает стартовый кодон (*) и стоп-кодоны, которые, возможно, указывают старт HCV ОРС. На фиг. 12 также указаны предположительно маленькие кодируемые пептиды (#), которые, возможно, играют роль в регуляции трансляции так же, как предположительно первая аминокислота предположительного полипептида (/) и нижележащие кодируемые аминокислоты.
Выделение и последовательность клона CA205a
Клон CA205a был выделен из лямбда-gt11 библиотеки (АТСС N 40394), используя синтетическую пробу, берущую начало из последовательности клона CA290a (фиг. 11). Последовательность пробы была:
5' TCA GAT CGT TGG TGG AGT TTA CTT GTT GCC 3'.
Последовательность HCV кДНК в CA205a, показанная на фиг. 13, перекрывается с кДНК последовательностями как клона ag30a, так и CA290a. Перекрывание этой последовательности с CA290a показано прерывистой линией выше последовательности (чертеж также демонстрирует предположительные аминокислоты, кодируемые этим фрагментом).
Как видно из HCV кДНК последовательностей, в клонах CA205a и ag30a предположительный HCV полипротеин, по-видимому, начинается в АТГ стартовом кодоне, HCV последовательности обоих клонов содержат в рамке два прилежащих стоп-кодона (ТГАТАГ) на сорок два нуклеотида выше этого АТГ. OPC, как кажется, начинается после этих стоп-кодонов и простирается по крайней мере на 8907 нуклеотидов (см. составную HCV кДНК, представленную на фиг. 18).
Выделение и последовательность клона 18g
Основываясь на последовательности клона ag30a (см. фиг. 12) и перекрывающегося клона из оригинальной библиотеки лямбда-gt11 (АТС N 40394) CA230a, была сделана синтетическая проба, имеющая следующую последовательность:
5' CCA TAG TGG TCT GCG GAA CCG GTG AGT ACA 3'.
Скрининг оригинальной лямбда-gt11 HCV кДНК библиотеки с пробой давал клон 18g, HCV кДНК последовательность которого показана на фиг. 14. Также на чертеже показано перекрывание с клоном ag30a и предположительные полипептиды, кодируемые в HCV кДНК.
кДНК в клоне 18g (C18g или 18g) перекрывается с клонами 30a и CA205a, описанными выше. Последовательность C18g также содержит район двойного стоп-кодона, наблюдаемый в клоне ag30a. Полинуклеотидный район выше этих стоп-кодонов предположительно представляет часть 5'-района HCV генома, который, возможно, содержит короткие ОРС и который можно подтвердить прямым сиквенсом очищенного HCV генома. Эти предположительные малые кодируемые петиды, возможно, играют роль регуляторную в трансляции. Район HCV генома выше района, представленного в C18g, может быть изолирован для анализа последовательности, используя по существу технологию, описанную в EPO publication N 318216 для выделения кДНК последовательностей, лежащих выше HCV кДНК последовательности в клоне 12f. По существу маленькие синтетические полинуклеотидные праймеры для обратной транскриптазы, которые основываются на последовательности C18g, синтезированы и используются для связывания соответствующей последовательности в HCV геномной РНК. Последовательности праймера проксимальны к известному 5'-концу C18g, но в значительной степени лежат ниже, чтобы позволить создать последовательности проб выше последовательностей праймеров. Используют стандартные методы праймирования и клонирования. Полученные кДНК библиотеки скринируют с последовательностями выше сайтов праймирования (как установлено из разъясненной последовательности C18). Геномную РНК HCV получают либо из плазмы, либо из образцов печени индивидуумов с NANBH. Так как HCV, по-видимому, является флавиподобным вирусом, 5'-конец генома может быть модифицирован "кэп" структурой. Известно, что геномы флавивирусов содержат 5-концевые "кэп" структуры (вирус желтой лихорадки, Rice и др. (1988), вирус лихорадки денге, Hahn и др. (1988), вирус японского энцефалита (1987)).
Выделение и последовательность клонов из бета-HCV кДНК библиотеки
Клоны, содержащие кДНК, представляющую 3'-концевой район HCV генома, были выделены из кДНК библиотеки, сконструированной из пула плазмы инфицированной шимпанзе, которую использовали для создания HCV кДНК лямбда-gt11 библиотеки (АТСС N 40394), описанной в EPO publication N 318216. Для того чтобы создать ДНК библиотеку, экстрагированная РНК из плазмы была снабжена "хвостом" поли A, используя поли(pA)-полимеразу, и синтезировали кДНК, используя олиго(дТ)12-18 в качестве праймера для обратной транскрипции. Полученный РНК:ДНК гибрид переваривали РНКазой H и превращали в двухцепочечную HCV кДНК. Полученную HCV кДНК клонировали в лямбда-gt10, используя по существу технологию, описанную Huynh (1985), что давало бета (или b) HCV кДНК библиотеку. Использованные процедуры были следующими.
Аликвоту (12 мл) плазмы обрабатывали протеиназой K и экстрагировали с равным объемом фенола, насыщенного 0,05 M трис-HCl, pH 7,5, 0,05% (объем/объем) бета-меркаптоэтанол, 0,1% (вес/объем) гидрокси, 1 мМ ЭДТА. Полученную водную фазу реэкстрагировали смесью фенола, после чего следовало 3 экстракции с 1:1 смесью, содержащей фенол и хлороформ:изоамиловый спирт (24:1), затем следовали 2 экстракции смесью хлороформа и изоамилового спирта (1:1). Вслед за сведением водной фазы к 200 мМ по NaCl нуклеиновые кислоты в водной фазе осаждали в течение ночи при -20oC 2,5 объемами холодного абсолютного спирта. Преципитаты собирали центрифугированием при 10000 об/мин в течение 40 минут, промывали 70% этанолом, содержащим 20 мМ NaCl, и 100% холодным этанолом, высушивали 5 минут в дессикаторе и растворяли в воде.
Выделенные из пула плазмы инфицированных шимпанзе нуклеиновые кислоты снабжались "хвостом" поли-pA, используя поли-A полимеразу в присутствии рибонуклеазного ингибитора из плаценты человека (HPRI) (закуплено у Amersham Corp. ), используя MS2 РНК в качестве носителя. Выделенные нуклеиновые кислоты, эквивалентные 2 мл плазмы, инкубировали в растворе, содержащем TMN (50 мМ трис-HCl, pH 7,9, 10 мМ MgCl2, 250 мМ NaCl, 2,5 мМ MnCl2, 2 мМ дитиотрейтол (ДТТ)). 40 микромоль альфа 32P АТФ, 20 единиц HPRI (Amersham Corp.) и около 9-10 единиц свободной от РНКаз поли-A полимеразы (BRL). Инкубацию проводили при 37oC 10 мин и останавливали реакцию ЭДТА (конечная концентарция около 250 мМ). Раствор экстрагировали равным объемом фенол-хлороформа и равным объемом хлороформа, и осаждали нуклеиновые кислоты в течение ночи при -20oC 2,5 объемами этанола в присутствии 200 мМ NaCl.
Выделение клона b5a
Бета HCV кДНК библиотеку скринировали гибридизацией, используя синтетическую пробу, которая имела последовательность, основанную на HCV кДНК последовательности клона 15e. Выделение клона 15e описано в EPO publication N 318216, и его последовательность показана на фиг. 3. Последовательность синтетической пробы была:
5' ATT GCG AGA TCT ACG GGG CCT GCT ACT CCA 3'.
Скрининг библиотеки дал клон бета-5a (b5a), который содержит HCV кДНК район приблизительно 1000 пар оснований. 5'-район этой кДНК перекрывается с клонами 35f, 19g, 26g и 15e (эти клоны описаны выше). Район между 3'-концевой поли-A последовательностью и 3'-последовательностью, который перекрывает клон 15e, содержит приблизительно 200 пар оснований. Этот клон позволяет идентифицировать район 3'-терминальной последовательности HCV генома.
Последовательность b5a содержится внутри последовательности HCV кДНК клона 16jh (описанного ниже). Более того, эта последовательность присутствует также в CC34a, выделенном из оригинальной лямбда-gt11 библиотеки (АТСС N 40394). (Оригинальная лямбда-gt11 библиотека называется здесь "C" библиотека).
Выделение и последовательность клонов, созданных амплификацией 3'-района генома HCV
Множественные кДНК клоны были созданы, содержащие нуклеотидные последовательности, берущие начало из 3'-района генома. Это было осуществлено путем амплификации района мишени генома полимеразной цепной реакцией, описанной Saiki и др. (1986) и Saiki и др. (1988), которую модифицировали как описано ниже. РНК HCV, которая была амплифицирована, была получена из оригинального пула плазмы инфицированной шимпанзе, которую использовали для создания HCV кДНК лямбда-gt11 библиотеки (АТСС N 40394), описанной в EPO publication N 318216. Выделение РНК HCV проводили как описано выше. К выделенной РНК добавляли на 3'-конце АТФ поли-A-полимеразой E. coli, как описано Sippel (1973), за исключением того, что изолированные из сывороток шимпанзе нуклеиновые кислоты были заменены на субстрат - нуклеиновую кислоту, РНК с поли-A была затем подвержена обратной транскрипции с получением кДНК с помощью обратной транскриптазы, используя олиго дТ-праймер-адаптер в значительной степени так, как описано Han (1987), за исключением того, что компоненты и последовательность праймер-адаптера была:
Результирующую кДНК подвергали амплификации путем PCR, используя два праймера: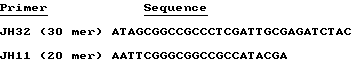
Праймер JH32 содержал 2o нуклеотидные последовательности, гибридизующиеся с 5'-концом района мишени в кДНК с определенной Tm 66oC. JH11 происходил из части олиго дТ-праймер-адаптера, таким образом он специфичен к 3'-концу кДНК при Tm 64oC. Оба праймера были сделаны так, чтобы иметь рестрикционный сайт для фермента рестрикции, NotI, на 5'-конце, для использования в последующем клонировании амплифицированной HCV кДНК.
Реакцию PCR проводили суспендируя кДНК и праймеры в 100 микролитрах реакционной смеси, содержащей четыре дезоксирибонуклеозид трифосфата, буферные соли и ионы металлов, и термостабильную ДНК-полимеразу, выделенную из Thermus aquaticus (Tag полимераза), которая есть в Perkin Elmer Cetus ките (N 801-0043 или N 801-0055). Реакцию PCR проводили в 35 циклов в ДНК-термальном амплификаторе Perkin Elmer Cetus. Каждый цикл состоял из 1,5-минутной стадии денатурации при 94oC, стадии отжига при 60oC, 2 мин, стадии элонгации праймера при 72oC, 3 мин. Продукты PCR подвергали Саузерн-блот анализу, используя 3o-нуклеотидную пробу JH34, последовательность которой основывалась на последовательности 3'-терминального района клона 15e. Последовательность JH34:
5' CTT GAT CTA CCT CCA ATC ATT CAA AGA CTC 3'.
Продукты PCR, детектированные HCV кДНК пробой, имеют разброс в размере от около 50 до около 400 пар нуклеотидов.
Для того чтобы клонировать амплифицированную HCV кДНК, PCR продукты расщепляли NotI и разделяли по размеру электрофорезом в полиакриламидном геле. ДНК больше чем 300 пар нуклеотидов (пн) клонировали в NotI сайт PUC18S. Вектор PUC18S конструируется включением NotI полилинкера, клонированного между сайтами EcoRI и SalI фиг. 18. Клоны скринировали на кДНК, используя JH34 пробу. Целый ряд политивных клонов было получено и секвенировано. Нуклеотидная последовательность HCV кДНК инсерции одного из этих клонов, 16 jh, и аминокислоты, кодируемые им, показаны на фиг. 15. На чертеже указана нуклеотидная гетерогенность, обнаруженная в последовательности HCV кДНК в клоне 16 jh по сравнению с другим клоном этого района.
Выделение и последовательность клона 6K
На основании последовательности клона 16 jh и клона b5a (см. выше) была сделана синтетическая проба, имеющая следующую последовательность:
5' TCT TCA ACT GGG CAG TAA GAA CAA AGC TCA 3'.
Скрининг оригинальной лямбда-gt11 HCV кДНК библиотеки (описанной в EPO publication N 318216) с пробой дал клоны
с частотой приблизительно 1 из 106, один их них был назван клон 6K (также названный C6K), HCV кДНК последовательность которого показана на фиг. 16. Также на чертеже показано перекрывание с клоном 16jh и предположительные полипептиды, кодируемые HCV кДНК. Информация о последовательности HCV кДНК в клоне 6K получена только с одной цепи. Информация об этом клоне дана ниже, где клон внесен в список как лямбда-gt11 C6K. Конформация C6K последовательности как часть ОРС, кодирующей HCV1 полипептид, получена при сиквенсе других перекрывающихся клонов.
Выделение и последовательность клона p131jh
Клон, содержащий последовательность из 3'-района HCV генома и который содержит в рамке стоп-кодон, был изолирован как описано выше для выделения клонов, образованных PCR амплификацией 3'-района генома, за исключением того, что РНК HCV1 была превращена в кДНК, используя олигонуклеотид
5' AAT TCG CGG CCG CCA TAC GAT TTA GGT GAC ACT ATA GAA T15 3'.
Эту кДНК затем амплифицировали реакцией PCR, используя праймеры
5' TTC GCG GCC GCT ACA GCG GGG GAG ACA T 3'
и
5' AAT TCG CGG CCG CCA TAC GA 3'.
После амплификации продукты PCR осаждали спермином, переваривали NotI и экстрагировали фенолом. Очищенные продукты клонировали в NotI сайт puc 18S и позитивные клоны отбирали, используя олигонуклеотид:
кДНК одного клона, обозначенного p131jh, показана на фиг. 17. Этот клон содержит в рамке стоп-кодон в большой рамке (OPC), содержащейся в HCV геноме.
Выделение и последовательность клона 5'-клон 32
Клон, содержащий последовательность из 5'-района HCV генома, выше последовательности клона b114a, был выделен и была определена нуклеотидная последовательность путем модификации метода выделения и определения последовательности клонов, созданных PCR амплификацией 3'-района генома, описанным в US N 456637, который вынесен в ссылки. В основном район мишени генома амплифицировали технологией PCR, описанной Saiki и др. (1986) и Saiki и др. (1988). HCV РНК, которая была амплифицирована, получена экстракцией человеческой сыворотки (US клинический изолят, HCV 27), используя холодный гаунидин тиоционатный метод, описанный Han и др. (1987). Экстрагированную РНК превращали в одноцепочечную кДНК обратной транскриптазой, используя праймер, JH94, который комплементарен нуклеотидам -250 по -223 генома HCV (см. фиг. 18). Последовательность JH94
5' CCT GCG GCC GCA CGA CAC TCA TAC TAA 3'.
Превращение одно- в двухцепочечную PCR кДНК было выполнено путем добавления к ДНК "хвоста" из приблизительно 20-50 дА остатков, используя терминальную дезоксинуклеотидил трансферазу (Sambrook и др. (1989), Molecular cloning) и репликацию полученной молекулы, используя следующий олиго-дТ праймер-адаптер, который содержит NotI сайт и sp6 промотор: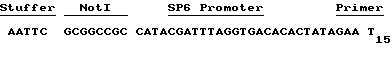
Полученную кДНК подвергали амплификации PCR, используя два праймера, JH94 (описанный выше) и JH11, который имеет следующую последовательность:
PCR реакцию проводили суспендируя кДНК и праймеры в 100 микролитрах реакционной смеси, содержащей четыре дезоксинуклеозид трифосфата, буферные соли и ионы металлов, и термостабильную ДНК-полимеразу, выделенную из thermus aquaticus (Taq-полимеразу), которая есть в Perkin Elmer Cetus ДНК термальном ките (N 801-0043 или N 801-0055). PCR реакцию выполняли за 35 циклов в термальном циклизаторе Perkin Elmer Cetus. Каждый цикл состоял из 1,5-минутной стадии денатурации при 94oC, стадии отжига при 60oC, 2 мин, и стадии элонгации праймера при 72oC, 3 мин.
Продукты PCR переваривали NotI и клонировали в puc 18S. Клоны, содержащие HCV нуклеотидные последовательности, были получены скринированием с пробой. Alex 90, которая происходила из -312 по -283 генома HCV1 и имеет последовательность:
5' ACC ATG AAT CAC TCC CCT GTG AGG AAC TAC 3'.
кДНК выделенных клонов секвенировали дидезокси-терминирующим методом (Sanger и др. (1977)). Последовательность кДНК одного из выделенных клонов, 5-клон 32, охватывает район нуклеотидов с -224 по -341 на фиг. 18.
Анализ нуклеотидной последовательности HCV кДНК показал, что реплика с HCV РНК цепи содержит ГЦ-богатую область, которая, возможно, способна формировать стабильную структуру шпильки: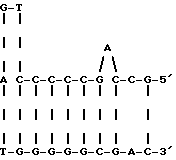
В этой структуре прерывистые линии указывают возможные водородные связи между комплементарными нуклеотидами.
Поиск в компьютерном банке данных Genebank обнаружил, что отсутствовали гомологичные последовательности из известных вирусных последовательностей. Таким образом, эта последовательность, возможно, уникальна на 5'-конце HCV генома.
Шпилечная структура может служить как сигнал узнавания транскриптазы и/или она может вносить вклад в стабильность РНК на 5'-конце.
Составленные HCV кДНК последовательности
HCV кДНК последовательность была собрана из серий перекрывающихся клонов, берущих начало из различных HCV кДНК библиотек, описанных здесь и в EPO publication N 318216. Клоны с фиг. 18 являются клонами 5'-32, b114a, 18g, ag30a, CA205a, CA290a, CA216a, pi14a, CA167b, CA156e, CA84a, CA59a, K9-1, 26j, 13i, 12f, 14i, 11b, 7f, 7e, 8h, 33c, 40b, 37b, 35, 36, 81, 32, 33b, 25c, 14c, 8f, 33f, 33g, 39c, 35f, 19g, 26g, 15e, b5a, 16jh, C6K and p131jh.
Методы выделения этих клонов так же, как их последовательности, здесь описаны и в EPO publication N 318216, которые вынесены здесь в ссылки. На фиг. 18 три штриха выше последовательности указывают положение предположительного метионинового кодона инициации.
Клон b114a перекрывается с клонами 18g, ag30a и CA205a, за исключением того, что клон b114a содержит дополнительно два нуклеотида выше последовательности в клоне 18g (т.е. 5'-CA). Эти два дополнительных нуклеотида включены в HCV геномную последовательность, показанную на фиг. 18.
Следует заметить, что хотя несколько описанных выше клона получено из других библиотек, чем оригинальная HCV кДНК лямбда-gt11 C библиотека (АТСС N 40394), эти клоны содержат кДНК последовательности, которые перекрываются с HCV кДНК последовательностями оригинальной библиотеки. Таким образом, по существу, вся HCV последовательность происходит из оригинальной лямбда-gt11 C библиотеки (АТСС N 40394), которую использовали для выделения первого HCV кДНК клона (5-1-1). Выделение клона 5-1-1 описано в EPO publication N 318216, который вынесен здесь в ссылки.
Показана также предположительная последовательность большого HCV полибелка, кодируемого в составной HCV1 кДНК. Первая аминокислота в последовательности - предположительный инициаторный метионин большой OPC. Вариантные аминокислоты, обусловленные гетерогенностью клонов, указаны выше последовательности. Так как лямбда-gt11 библиотека была создана из сыворотки, полученной из одного индивидуума (см. EPO publication N 318216), результаты предполагают, что вариантные вирусные последовательности (как нуклеотидные, так и аминокислотные) присутствуют в этом индивидууме.
Исследование составной HCV кДНК последовательности показывает, что помимо большой OPC имеет целый ряд OPC выше OPC, кодирующей полибелок, и внутри последовательности, кодирующей полибелок, есть большое количество меньших OPC в двух других трансляционных рамках. OPC выше HCV полибелка показаны в таблице 1.
Рамка считывания, позиция и размер ОРС ниже последовательности, кодирующей предположительно инициаторный МЕТ полибелка, показаны в таблице 2. Большой полибелок - тот, что транслируется с рамки считывания 2.
В дополнение к вышесказанному исследование последовательности, которая комплементарна геномной цепи HCV РНК, также показало, что она содержит несколько маленьких OPC. Одна из этих OPC, которая комплементарна нуклеотидам с -341 по +837 в HCV РНК последовательности, кодирует полипептид 385 аминокислот.
Сравнение последовательностей 5'-районов, полученных из HCV изолятов из разных географических областей
Нуклеотидные последовательности 5'-районов HCV изолятов из США (HCV18, HCV27), из Италии (HCVI1, HCVI24) и Кореи (HCVK1) сравнили.
Выделение HCV кДНК последовательностей по существу проводилось, как описано выше, для выделения 5'-клона 32, за исключением следующего. Экстрагированную РНК обратно транскрибировали в кДНК, используя в качестве праймеров либо JH51, либо r16, которые комплементарны HCV нуклеотидам с -90 по -73 и с 366 по 383 соответственно. Последовательности этих праймеров следующие
Амплификацию HCV дцДНК делали методом PCR, используя JH93 и JH52 как 5'- и 3'-праймеры соответственно, HCV последовательность в JH93 происходила из HCV нуклеотидов с -317 по -296, в JH52 с -93 по -117, номер нуклеотида указан в скобках внизу последовательности. В JH52 подчеркнутый динуклеотид был мутирован для создания NotI сайта. Последовательности этих праймеров следующие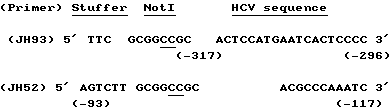
После амплификации продукты PCR расщепляют NotI и клонируют в puc 18S. HCV кДНК секвенировали или непосредственно после амплификации PCR или, альтернативно, секвенировали клонированные HCV кДНК путем наращивания праймера или дидезокси методом. Наращивание праймера и дидезокси метод сиквенса выполнялись как описано выше для последовательности 5'-клона 32.
PCR метод для прямого сиквенса использовал Alex 90 (см. выше последовательность) как 5'-праймер и r25 как 3'-праймер. Alex 90 происходил из HCV нуклеотидов с -312 по -283 и r25 происходил из нуклеотидов с 365 по 342 (см. фиг. 18). Последовательность r25:
5' ACC TTA CCC AAA TTG CGC GAC CTA 3'.
Сравнение последовательностей 5' района 'HCV27, HCVK1, HCV1 24, HCVI1 и HCV18 с последовательностью прототипа HCV, HCV1 показало следующее. Исследованный район-5' высококонсервативен среди 5'HCV изолятов. По-видимому, последовательности эти идентичны, за исключением одного нуклеотида, делетированного в положении 171 в HCV 124, и двусмысленноcть в четырех нуклеотидах в положениях -222 по -219 в изоляте HCVK1.
Высокие уровни консервативности последовательности в этом районе могут отражать роль этого района в вирусной репликации, и/или транскрипции, и/или трансляции.
Вариации последовательности HCV изолятов из различных индивидуумов
Изоляты HCV, которые содержат последовательности, которые происходят от CDC/HCV1, были идентифицированы в человеческих индивидуумах, некоторые из которых были серологически позитивны на анти-C100-3 антитела (EC10 был антитело негативным). Идентификация этих новых изолятов проводилась путем клонирования и секвенирования сегментов HCV генома, которые были амплифицированы технологией PCR, используя CDC/HC1 последовательности. Амплификацию выполняли по существу на основании HCV/cPCR метода. Этот метод использует праймеры и пробы, основанные на кДНК последовательностях, здесь описанных. Первая стадия метода - синтез кДНК, либо HCV генома, либо его репликативного интермедиата, используя обратную транскриптазу. После синтеза HCV кДНК и перед амплификацией РНК образца деградируют при помощи технологий, известных в науке. Желаемый сегмент HCV кДНК затем амплифицируют при использовании подходящих праймеров. Амплифицированные последовательности клонируют и клоны, содержащие амплифицированные последовательности, детектируют с пробой, которая комплементарна последовательности, лежащей между праймерами, но которая не перекрывает праймеры.
HCV изоляты, выделенные из людей в США
Образцы крови, которые использовали как источник HCV варионов, были получены из American Red Cross в Charlotte, North Carolina, и из Community Blood Center of Kansas, Kansas City, Missous. Образцы были прокскринированы на антитела к HCV C100-3 антигену, используя ELISA анализ? как описано в EPO publication N 318216, и подвержены дополнительному Вестерн-анализу, используя поликлональную козлиную античеловеческую HPR для измерения антиHCV-антител. Два образца, #23 и #27, из American Red Cross и из Community Blood Center of Kansas соответственно, как определяли, являются HCV позитивными в этих анализах.
Вирусные частицы, присутствовавшие в сыворотке этих образцов, выделяли ультрацентрифугированием при условиях, описанных Bradley и др. (1985). РНК экстрагировали из частиц перевариванием с протеиназой К и ДДС в конечной концентрации 10 микрограмм/мл протеиназы К и 0,1% ДДС, переваривание проводили в течение 1 часа при 37oC. Вирусную РНК затем очищали экстракцией хлороформ-фенолом, как описано в EPO publication N 318216.
HCV РНК в препаратах РНК обратно транскрибировали в кДНК по существу, как описано в EPO publication N 318216, за исключением того, что олигонуклеотид JHC 7, который соответствует кДНК последовательности 1958-1939 и который имеет следующую последовательность, использовали как праймер реакции обратной транскрипции.
JHC 7: CCA GCG GTG GCC TGG TAT TG.
После синтеза обоих цепей кДНК полученную кДНК затем амплифицировали PCR методом, по существу, как описано выше, для выделения клонов, созданных PCR амплификацией, за исключением того, что использованные нуклеотидные праймеры, т. е. JHC6 и ALX80 были предназначены для амплификации 1080 нуклеотидного сегмента HCV генома из CDC/HCV1 нуклеотидов с 673 по 1751. Праймеры, кроме того, были предназначены для введения сайта рестрикции в 3'-конец PCR продукта и тупого 5'-конца. Последовательности праймеров:
ALX 80: TTT GGG TAA GGT CAT CGA TAC CCT TAC GTG;
и
JHC 6: ATA TGC GGC CGC CTT CCG TTG GCA TAA.
ALX 80 соответствует нуклеотидам 673-702 CDC/HCV1 последовательности, JHC6 соответствует нуклеотидам 1752-1738 HCV1 (в дополнение, имеются 12 экстрануклеотидов, которые кодируют NotI сайт). Обозначение нуклеотидов в JHC 6, т.е. номер из положения, указывает место в антисмысловой цепи.
После PCR амплификации с вышеописанных праймеров тупые концы превращали в NotI сайт следующим образом. Гомополимерный хвост из 15 дГ присоединяли к PCR продукту, используя терминальную дезоксинуклеотид трансферазу, и продукты вновь подвергали амплификации PCR, используя в качестве праймеров JHC6 и JHC13. Последний праймер, JHC13, последовательность которого следует ниже, сделан, чтобы внести NotI сайт в дополнение к Sp6 фаговому промотору. (Sp6 промотор описан в Genetic engineering, J.Setlow Ed. (1988)).
JHC 13: AAT TCG CGG CCG CCA TAC GAT TTA GGT GAC ACT ATA GAA CCC CCC CCC CCC CCC.
Для того чтобы клонировать амплифицированную HCV кДНК, продукты PCR расщепляли NotI, осаждали спермином для удаления свободных олигонуклеотидов (Hoopes и др. 1981), и клонировали в NotI сайте puc 18S (см. раздел IV.A. 34). HCV кДНК трех клонов, происходившая из каждого HCV изолята, подвергали сиквенсовому анализу. Анализ, по существу, проводился в соответствии с методом, описанным Chem и Seeburg (1985).
Последовательности консенсуса клонов, берущих начало из HCV образца 23, показаны на фиг. 39.
Фиг. 34 и 35 демонстрируют сравнение построчно позитивной цепи нуклеотидной последовательности (фиг. 34) и предположительных аминокислотных последовательностей (фиг. 35) образцов 23, 27 и HCV1. Аминокислотная последовательность HCV1 на фиг. 34 представляет аминокислотные номера 129-467 HCV полибелка, кодируемого большой OPC HCV геномной РНК. Исследование фиг. 39 показывает, что есть вариации в последовательностях трех выделенных клонов. Вариации последовательностей на нуклеотидном уровне и на аминокислотном уровне суммированы в таблице 3. В таблице 3 полипептид, обозначенный S и NS1, представляет аминокислоты номеров с 130 по ~ 380, и 380 по ~ 470 соответственно. Нумерация идет от предположительного инициаторного метионина. Терминология S и NS1 основана на позициях последовательностей, кодирующих полипептиды, используя модель флавивирусов. Как обсуждалось выше, однако, недавнее свидетельство предлагает, что нет полной корреляции между HCV и флавивирусами в отношении вирусных полипептидных доменов, особенно в предположительных E/NS1 доменах. Действительно, HCV полипептиды и кодирующие их домены могут демонстрировать существенное отклонение от флавивирусной модели.
Хотя есть отлонения в нововыделенных HCV последовательностях, клонированные последовательности из образцов 23 и 27 (названных HCV 23 и HCV 27) каждый содержит 1019 нуклеотидов, указывая на отсутствие делеции и других мутантов в этом районе в изолированных клонах. Последовательности на фиг. 34 и 35 также показывают, что выделенные последовательности не перестроены в этом районе.
Сравнение последовательностей консенсусов для HCV1 и для других изолятов HCV суммировано в таблице 3. Вариации последовательностей изолята шимпанзе HCV1 и HCV, изолированных из людей, почти те же, что и наблюдаемые между HCV из людей.
Интересно, что вариации последовательностей из двух предположительных доменов неодинаковы. Последовательность предположительного S района, по-видимому, относительно константна и случайно разбросана по геному. В противоположность этому предположительный NS1 район имеет более высокую степень вариабельности, чем вся последовательность и вариации, по-видимому, есть в гипервариабельном кармане около 28 аминокислот, которые локализуются около 70 аминокислот ниже предположительного N-конца предположительного полибелка.
Хотя можно утверждать, что обнаруженные вариации были введены во время процесса амплификации, не похоже, что все вариации произошли от этого. Было оценено, что Tag-полимераза вводит ошибки в последовательность приблизительно одну на 10 тысяч пар нуклеотидов ДНК матрицы на цикл (Saiki и др., 1988). На основании этой оценки до 7 ошибок могло быть введено во время PCR амплификации 119 пн фрагмента ДНК. Однако три субклона HCV 23 и HCV 27 давали 29 и 14 нуклеотидов вариантных соответственно. Нижеследующие данные наводят на мысль, что эти вариации случаются естественным путем. Около 60% нуклеотидных замен - молчащие мутации, которые не изменяют аминокислотную последовательность. Вариации, введенные Tag-полимеразой, во время PCR амплификации, как следовало бы ожидать, происходят случайно, однако результаты показывают, что вариантные последовательности образуют кластер по крайней мере в одном специфическом районе. Более того, последовательности консенсуса происходили при сиквенсе множества различных клонов из PCR амплифицированных продуктов.
HCV изоляты из пациентов в Италии и в США
Сегменты HCV РНК, присутствующие в различных изолятах, амплифицировали методом HCV/c PCR. Эти сегменты охватывают район ~ 0,6 тпн по ~ 1,6 тпн ниже от метионина, кодирующего старткодон предположительного HCV полибелка. Изоляты из биологических образцов, полученных от HCV инфицированных индивидуумов. Более подробно, изолят HCV # 18 из человеческой плазмы индивидуума в США, EC1 и EC10 из биопсии печени итальянского пациента, и Th из фракции периферической крови мононуклеоцитов африканского пациента. Сравнимые сегменты HCV РНК изолированы из шимпанзе.
РНК экстрагировали из образцов человеческой плазмы, используя фенол: CHCl3:изоамиловый спирт экстракцию. Или 0,1 мл, или 0,01 мл плазмы разводили до конечного объема 1,0 мл TENB протеиназа К/ДДС раствором (0,05 М трис-HCl, pH 8,0, 0,001 М ЭДТА, 0,1 М NaCl, 1 мг/мл протеиназы K и 0,5% ДДС), содержащим 10-40 микрограмм/мл полиаденилиновой кислоты и инкубировали при 37oC 60 минут. После этой протеиназной обработки фракции полученной плазмы депротеинизировали эктракцией ТE (50 мМ Трис-HCl, pH 8,0, 1 мМ ЭДТА) насыщенным фенолом, pH 6,5. Фенольную фазу отделяли центрифугированием и реэкстрагировали TENB, содержащим 0,1% ДДС. Полученные водные фазы из каждой экстракции объединяли и дважды экстрагировали равным объемом фенол/хлороформ/изоамиловый спирт (1:1 99:1) и затем дважды с равным объемом 99: 1 смеси хлороформ/изоамиловый спирт. Затем следовало разделение на фазы центрифугированием и водную фазу приводили к конечной концентрации 0,2 M Na ацетат, и осаждали нуклеиновые кислоты добавлением двух объемов этанола. Осажденные нуклеиновые кислоты выделяли ультрацентрифугированием в SW 41 роторе при 38 тыс. об/мин в течение 60 минут при 4oC или в микроцентрифуге 10 минут при 10 тыс. об/мин.
РНК, экстрагированную из биопсии печени, поставляли от Dr. F.Bonino, Ospedale Maggiore di S.Giovanni Battista, Torino, Italy.
Фракцию мононуклеоцитов получали седиментацией индивидуальной аликвоты крови через фиколл-Paque® (Pharmacia Corp), используя указания производителя. Тотальную РНК экстрагировали из фракции, используя гуанидин тиоцинатную процедуру, описанную в EPO publication N 318216 (см. также Choo и др., 1989).
Синтез HCV кДНК из образцов выполняли, используя обратную транскриптазу и праймеры, происходящие из клонов 156e и K91. Эти праймеры, которые являются антисмысловыми по отношению к геномной РНК, имеют следующие последовательности
156e 16B: 5' CGA CAA GAA AGA CAG A 3'
и
K91/16B 5' CGT TGG CAT AAC TGA T 3'.
Вслед за этанольной преципитацией осажденную РНК или фракцию нуклеиновых кислот высушивали и ресуспендировали в обработанной ДЕРС дистиллированной воде. Вторичные структуры нуклеиновых кислот разрушались при нагревании до 65oC 10 минут и образцы сразу охлаждали во льду. кДНК синтезировали, используя от 1 до 3 микрограмм тотальной РНК из печени и из нуклеиновых кислот, или РНК, экстрагированных из от 10 до 100 микролитров плазмы. В синтезе использовали обратную транскриптазу и проводили реакцию в 25 микролитрах, используя описание, выпускаемое производителем, BRL. Все реакционные смеси для синтеза кДНК содержали 23 единицы ингибитора РНКаз, RNASINm (Fisher/promega). После синтеза кДНК реакционные смеси разводили водой, кипятили 10 минут и быстро охлаждали во льду.
Каждый набор образцов подвергали двум раундам амплификации. Праймеры для реакций отбирали таким образом, чтобы амплифицировать районы, обозначенные "EnvL" и "EnvR". "EnvL" район содержит нуклеотиды 669-1243 и предположительные аминокислоты 117-308, "EnvR" район содержит нуклеотиды 1215-1629 и кодирует предположительные аминокислоты 300-408 (предположительные аминокислоты нумеруются, начиная с предположительного метионинового инициаторного кодона). Взаимосвязь этих районов по отношению к предположительному полибелку, кодируемому HCV кДНК, и полипептидам, кодируемым по модели флавивирусов, показана на фиг. 40.
Праймеры для первого раунда PCR реакций происходили из HCV кДНК последовательностей либо клона ag30a, клона 156e, либо клона K9-1. Используемые праймеры для амплификации района были 156e16B (показан выше) и ag30a16A для смысловой цепи. Амплификация EnvR района использовала праймер K92/16B (показан выше) и 156e16a для смысловой цепи. Последовательности праймеров смысловой цепи следующие
For EnvL, ag30a16A: 5' CTC TAT GGC AAT GAG G 3'
и
For EnvR, 156e16A: 5' AGC TTC GAC CTC ACA T 3'.
Реакции PCR выполнялись в значительной степени согласно указаниям производителя (Cetus-Perkin-Elmer), за исключением того, что добавляли 1 микрограмм РНКазы A. Реакции проводили в конечном объеме 100 микролитров. PCR выполняли за 30 циклов, используя режим 94oC (1 мин), 37oC (2 мин) и 72oC (3 мин) с 7-минутной элонгацией при 72oC в последнем цикле. Образцы затем экстрагировали фенол:CHCl3, осаждали этанолом 2 раза, суспендировали в 10 мМ трис-HCl, pH 8,0 и концентрировали, используя Центрикон-30 (Amicon) фильтрацию. Эта процедура по существу удаляет олигонуклеотиды размером менее 30 нуклеотидов, таким образом удаляются праймеры из первого раунда PCR амплификации.
Центрикон-30 сконцентрированные образцы затем подвергали второму раунду PCR амплификации, используя пробы, полученные из клонов 202a и 156e для EnvL района и из 156e и 59a для EnvR района. Праймеры для амплификации EnvL района имеют следующие последовательности
202aEnv41a: 5' CTT GAA TTC GCA ATT TGG GTA AGG TCA TCG ATA CCC TTA CG 3'
и
156e38B': 5' CTT GAA TTC GAT AGA GCA ATT GCA ACC TTG CGT CGT CC 3'.
Праймеры для амплификации EnvR района в РНК, происходящей из людей, имеют следующие последовательности
156e38A': 5' CTT GAA TTC GGA CGA CGC AAG GTT GCA ATT GCT CTA TC 3'
и
59aEnv39C: 5' CTT GAA TTC CAG CCG GTG TTG AGG CTA TCA TTG CAG TTC 3'.
Амплификацию PCR проводили за 35 циклов, используя режим 94oC (1 мин), 60oC (1 мин), и 72oC (2 мин) с 7-мин. элонгацией при 72oC в последнем цикле. Образцы далее экстрагировали фенол:CHCl3, осаждали два раза и рестрицировали EcoRI. Продукты PCR реакции анализировали разделением их электрофорезом в 6% полиакриламидных гелях. ДНК приблизительно оцененной длины ожидаемого PCR продукта электроэлюировали из гелей и субклонировали либо в pGEM-4 плазмидном векторе, либо в лямбда-gt11. Продукт ожидаемого размера для EnvL и EnvR после первого раунда амплификации 615 пн и 683 пн соответственно, после второго раунда амплификации размеры ожидаемого продукта для EnvL и EnvR 414 пн и 575 пн соответственно. Плазмиды, содержащие амплифицированные продукты, использовали для трансформации клеток хозяина, pGEM-4 плазмида использовалась для трансформации DH5-альфа, и лямбда-gt11 использовалась для трансформации C600 дельта-HFL. Клоны трансформированных клеток, которые либо гибридизовали с подходящими HCV пробами (описанных ниже), либо те, которые инсертировали правильного размера. Инсерции были затем клонированы в М13 и секвенированы.
Пробы для всех HCV/cPCR продуктов состояли из 32P-меченых секций HCV кДНК, приготовленных PCR амплификацией района клона 216 (используя CA216a16A и 216a16B как праймеры) и клона 84 (используя CA84a16A и CA84a16B или CA84a16C как праймеры), 32P включали в продукты PCR ник-трансляцией. Пробы для первого и второго раунда EnvL амплификации были из клона 216. Проба для первого раунда EnvR амплификации были из 84 (т.е. CA84a16A и CA84a16B), для второго раунда EnvL амплификации были CA84a16A и CA84a16C. Эти пробы не перекрываются с праймерами, используемыми в реакциях HCV/cPCR. Последовательности праймеров для PCR амплификации проб приводятся в таблице 4.
Информация о последовательности вариантов в районе EnvL была получена из 3 клонов из HCT # 18, 2 клонов из TH, 3 клонов их EC1 им HCV1 клонов, описанных в EPO publication N 318216 и выше. Сравнение составной нуклеотидной последовательности каждого изолята, происходящего из этих клонов, показано на фиг. 41. На чертеже каждая последовательность показана от 5' к 3' для смысловой цепи EnvL района и эти последовательности были совмещены построчно. Вертикальные линии и заглавные буквы указывают гомологию последовательности, отсутствие линии и прописные буквы указывают отсутствие гомологии. Последовательности, показанные не в строчках, следующие: строчка 1, Thorn; строчка 2, EC1; строчка 3, HCT # 18; линия 4, HCV1.
Информация о последовательности вариантов в EnvR районе получена из двух клонов EC10 и из HCV1 клонов, описанных в EPO publication N 318216 и выше. Сравнение нуклеотидных последовательностей EC10 (клон 2) и составных HCV 1 последовательностей показано на фиг. 42, каждая последовательность представлена 5' и 3' для смысловой цепи EnvR района и последовательности совмещены построчно. Двойные штрихи между последовательностями указывают гомологию последовательностей.
Сравнение аминокислотных последовательностей, кодирующих EnvL (аминокислоты # 117-308) и EnvR район (аминокислоты # 300-438), для каждого изолята показано на фиг. 43 и 44 соответственно. В эти чертежи включены последовательности изолятов JH23 и JH27, описанных выше. Также указаны последовательности из японского изолята, эти последовательности поставляли от Dr. Miyamura, Japan. На чертежах дана аминокислотная последовательность для этого района во всей полноте для HCV1 и указаны негомологичные аминокислоты в различных изолятах.
Как показано на фиг. 43, в EnvL районе в целом около 93% гомология между HCV1 и другими изолятами. HCT18, Th и EC1 имеют около 97% гомологию с HCV1, JH23 и JH27 имеют около 96% и 95% гомологию соответственно с HCV1. Фиг. 44 показывает, что гомологий в EnvR районе значительно меньше, чем в EnvL районе, более того, кажется, что один субрайон является гипервариабельным (т.е. аминокислоты 383-405). Эти данные суммируются в таблице 5.
Детекция позитивной и негативной цепи 5'-HCV РНК в сыворотке
РНК HCV 27, изолированная из сыворотки, анализировали на присутствие позитивной и негативной цепей, используя метод PCR. PCR метод выполнялся в значительной мере как описано выше, за исключением следующего. Экстрагированную HCV27 РНК обратно транскрибировали в одноцепочечную кДНК, используя в качестве праймера либо Alex 90, либо JH52 (см. выше для последовательностей). Последовательность Alex 90 спаривается с ней в положениях -312 по -283 позитивной цепи HCV РНК, в то время как JH52 спаривается с нуклеотидами -117 по -93 негативной цепи. Полученные одноцепочечные HCV кДНК каждую отдельно амплифицировали PCR, используя Alex 90 и JH52. Обнаружение амплифицированных продуктов проводили Саузерн блоттингом, используя Alex 89 в качестве пробы. Alex 89 спаривается с нуклеотидами номеров с -203 по -175 HCV РНК. Последовательность Alex 89:
5' CCA TAG TGG TCT GCG GAA CCG GTG AGT ACA 3'.
Анализ показал, что этим методом сигналы амплифицированных продуктов обеих цепей РНК были равной интенсивности. Эти результаты предлагают, что HCV РНК в 5' районе, возможно, существует как двухцепочечная РНК.
Пробы для гибридизации по типу "сэндвича" для HCV
Этот пример приводит наборы меченых и захватывающих проб, полезных для детекции HCV РНК в биологических образцах, используя по существу анализ, описанный в US patent N 4868105. Этот метод является гибридизацией по типу "сэндвича" в водной фазе, при котором используются как захватывающая, так и меченая пробы, которые гибридизуются с последовательностями мишени в нуклеиновой кислоте аналита. При скрининге биологических образцов на HCV используемые пробы связываются с консервативными районами HCV генома, и HCV связывающиеся районы отбираются по их уникальности в HCV геноме. Районы, которые связываются со связывающим партнером захватывающей пробы или частью меченой пробы, которая связывается с меченым мотивом (или с амплифицированным мультимером, если описанный метод в EPO publication N 317077 использован) отбирают такие, что они не связываются с какой-либо из известных последовательностей в банке данных или в HCV, и которые имеют подходящий Г+Ц состав, чтобы допустить образование стабильных дуплексов с их комплементами при селективных условиях. Захватывающая и меченая пробы есть в наборах 1 - 3 (см. в конце описания) и пробы одного набора не перемешивают с пробами другого набора. Эти пробы состоят из HCV последовательностей кодирующей цепи последовательности прототипа HCV кДНК, показанной на фиг. 18. В вышеперечисленных наборах каждая проба содержит в дополнение к последовательностям, комплементарным HCV последовательностям, следующую последовательность ниже HCV последовательности (т.е. на 3-конце): 5' CTT CTT TGG AGA AAG TGG TG3'. Эта последовательность, общая для каждой захватывающей пробы, комплементарна последовательности связывающего партнера(ам), так что после гибридизации дуплекс можно захватить посредством его фиксации на твердой фазе.
Также в каждом наборе каждая меченая проба содержит в дополнение к последовательностям, комплементарным HCV последовательностям, следующую последовательность ниже последовательности HCV: 5' TTA GGC ATA GGA CCC GTG TC3'.
Если используют метод, описанный в EPO publication N 317077, последовательность, общая для каждой меченой пробы, комплементарна последовательности мультимера, чтобы допустить образование гибридных дуплексов с этим мультимером.
Последовательности проб вышеперечисленных наборов показаны на фиг. 19.
Детекция HCV полинуклеотидных последовательностей, используя PCR амплификацию
В общеупотребительном методе амплификации HCV РНК с PCR предполагают, что РНК цепь - вирионная или mРНК цепь, которая является "смысловой" цепью. Однако также представляется возможным, что репликативные интермедиатные формы можно также обнаружить, которые будут "антисмысловыми", в этом случае праймер будет "смысловым". Смысловая цепь РНК, содержащая район мишени, гибридизуется с антисмысловым праймером, который праймирует синтез репликативной цепи, содержащей мишень. кДНК на РНК матрице синтезируется праймер- и образецзависимой обратной транскриптазой. кДНК в полученном РНК:ДНК гибриде освобождается денатурацией и обработкой РНКазой. Праймеры отжигаются с кДНК и наращиваются праймер- и матрицезависимой ДНК-полимеразой. Продукты денатурируют, вновь отжигают с праймерами и проводят второй раунд синтеза. Некоторое количество циклов проводят до появления амплифицированного продукта, содержащего район мишени в желаемом количестве, которое по крайней мере соответствует детектируемому уровню.
Детекция амплифицированных HCV последовательностей нуклеиновых кислот, происходящих от HCV последовательностей нуклеиновых кислот в печени и образцах плазмы из шимпанзе с NANBH
HCV нуклеиновые кислоты, присутствующие в печени и плазме шимпанзе с NANBH и не присутствующие в контрольных шимпанзе, амплифицировали, используя по существу полимеразную цепную реакцию, описанную (PCR) Saiki и др. (1986). Олигонуклеотиды-праймеры происходили из HCV кДНК последовательностей клона 81 (фиг. 21) или клонов 36 (фиг. 22) и 37b (фиг. 24). Амплифицированные последовательности детектировали гель электрофорезом и модифицированным методом Саузерн блоттинга, используя в качестве проб подходящий кДНК олигомер или ник-транслированную кДНК последовательность с последовательностью из этого района между, но не включая два праймера. Образцы РНК, содержащие последовательности HCV, чтобы исследовать их в системе амплификации, были выделены из биопсий печени трех шимпанзе с NANBH и из двух контрольных шимпанзе. Выделение фракции полиA + РНК проводилось гуанидин тиоцианатной процедурой, описанной Maniatis и др. (1982).
Образцы РНК, которые были исследованы системой амплификации, были также выделены из плазм двух шимпанзе с NANBH и из одной контрольной шимпанзе так же, как и из пула плазмы контрольных шимпанзе. Одна инфицированная шимпанзе имела титр, равный или больше 106 CID/мл, и другая инфицированная шимпанзе имела титр, равный или больше чем 105 CID/мл.
Нуклеиновые кислоты экстрагировали из плазмы следующим образом. Либо 0,1 мл, либо 0,01 мл плазмы разбавляли до конечного объема 1,0 мл TENB (протеиназа K/раствор ДДС/0,05 M трис-HCl, pH 8,0, 0,01 М ЭДТА, 0,1 М NaCl, 1 мг/мл протеиназа K и 0,5% ДДС), содержащим 10 микрограмм/мл полиадениловую кислоту, и инкубировали при 37oC 60 минут. После этой обработки протеиназой K полученные фракции плазмы депротеинизировали экстракцией ТЕ (10,0 мМ трис-HCl, pH 8,0, 1 мМ ЭДТА) насыщенным фенолом. Фенольную фазу отделяли центрифугированием и реэкстрагировали TENB, содержащим 0,1% ДДС. Полученные водные фазы из каждой экстракции собирали и экстрагировали дважды равным объемом фенол/хлороформ/изоамиловый спирт (1:1 (99:1)), и затем дважды с равным объемом 99: 1 смеси хлороформ/изоамиловый спирт. После разделения фаз центрифугированием водную фазу приводили к конечной концентрации 0,2 М по Na ацетату и осаждали нуклеиновые кислоты добавлением двух объемов этанола. Осажденные нуклеиновые кислоты собирали ультрацентрифугированием в SW 41 роторе при 38 тыс. об/мин в течение 60 минут при 4oC.
В дополнение к вышесказанному высокий титр плазмы шимпанзе и контрольные плазмы пула альтернативно экстрагировали 50 микрограммами поли-A носителя при помощи процедуры Chomcyrski и (Sacchi (1987)). Эта процедура использовала гуанидин тиоционатную экстракцию. РНК откидывали центрифугированием при 1000 об/мин 10 минут при 4oC в центрифуге Эппендорф.
В двух случаях перед синтезом кДНК в реакции PCR нуклеиновые кислоты, экстрагированные из плазмы протеиназой К/ДДС/фенольным методом, в дальнейшем очищали связыванием и элюцией с S и S-колонок. Эта процедура проводилась в соответствии с указаниями Elutip-R производителя.
кДНК, использованная в качестве матрицы для реакции PCR, происходила из нуклеиновых кислот (или тотальные нуклеиновые кислоты, или РНК), приготовленных как описано выше. После осаждения этанолом преципитировавшие нуклеиновые кислоты высушивали и ресуспендировали в обработанной DEPC дистиллированной воде. Вторичные структуры нуклеиновых кислот разрушались прогреванием при 65oC в течение 10 минут и образцы сразу охлаждали во льду. кДНК синтезировала, используя от 1 до 3 микрограмм тотальной РНК шимпанзе из печени или микролитры плазмы. При синтезе использовали обратную транскриптазу и он проводился в 25 микролитрах реакционной смеси, используя описание, специфическое для производителя, BRL. Праймеры для синтеза кДНК были те, что также использовались в реакции PCR, описанной ниже. Все реакционные смеси для кДНК синтеза содержали 23 единицы ингибитора РНКаз, RNASINm (Fisher/promega). После синтеза кДНК реакционные смеси разбавляли водой, кипятили 10 минут и быстро охлаждали во льду.
Реакции PCR выполнялись по существу в соответствии с указаниями производителя (Cetus-Perkin-Elmer) за исключением добавления 1 микрограмма РНКазы A. Реакции проводились в конечном объеме 100 микролитров. PCR выполняли за 35 циклов, используя режим 37oC (2 мин), 72oC (3 мин) и 94oC (1 мин).
Праймеры для кДНК синтеза и для реакций PCR происходили из HCV кДНК последовательностей либо клона 81, клона 36, либо клона 37b (HCV кДНК последовательности клонов 81, 36 и 37b показаны на фиг. 21, 22 и 23 соответственно).
Последовательности двух 16-мерных праймеров, берущих начало от клона 81, были:
5' CAA TCA TAC CTG ACA G 3'
и
5' GAT AAC CTC TGC CTG A 3'.
Последовательность праймера из клона 36 была
5' GCA TGT CAT GAT GTAT 3'.
Последовательность праймера из клона 37b была:
5' ACA ATA CGT GTG TCA C 3'.
В реакциях PCR пары праймеров состоят либо из двух 16-меров, происходящих из клона 81, либо 16-меров из клона 36 и 16-мера из клона 37b.
Продукты PCR реакции анализировали разделением продуктов электрофорезом в щелочном геле, после чего следовал Саузерн блотинг и обнаружение амплифицированных HCV-кДНК последовательностей с 32P-меченой внутренней олигонуклеотидной пробой, полученной из района HCV кДНК, который не перекрывается с праймерами. Смеси из PCR реакции экстрагировали фенол/хлороформом и осаждали нуклеиновые кислоты из водной фазы солью и этанолом. Осажденные нуклеиновые кислоты собирали центрифугированием и растворяли в дистиллированной воде. Аликвоты образцов подвергали электрофорезу в 1,8% щелочном агарозном геле. Одноцепочечную ДНК размером 60, 108 и 161 нуклеотидов прогоняли совместно на гелях как маркеры молекулярного веса. После электрофореза ДНК в гелях переносили на Biorad Zeta probem бумагу. Условия предгибридизации и гибридизации и отмывки были те, что специфичны у производителя.
Пробы, используемые для детекции гибридизацией амплифицированных последовательностей HCV кДНК были следующие. Когда пара PCR праймеров происходила из клона 81, проба была 108-мер с последовательностью, соответствующей последовательности, которая локализована в районе между последовательностями двух праймеров. Когда пара PCR праймеров происходила из клона 36 и 37b, проба была ник-транслированной HCV кДНК инсерцией, происходящей из клона 35, нуклеотидная последовательность которого показана на фиг. 31. Праймеры происходили из нуклеотидов 155-170 клона инсерции 37 и 206-268 из клона инсерции 36. 3'-конец HCV кДНК инсерции в клоне 35 перекрывает нуклеотиды 1-186 инсерции в клоне 36, и 5'-конец клона инсерции 35 перекрывает нуклеотиды 207-269 инсерции клона 37b. (Сравните фиг. 23, 34 и 24). Таким образом, кДНК инсерция клона 35 охватывает часть района между последовательностями от клона 36 и 37b происходящих праймеров и используется как проба для амплифицированых последовательностей, которые включают эти праймеры.
Анализ РНК из образцов печени проводился в соответствии с вышеприведенной процедурой, используя как наборы праймеров, так и проб. ДНК из печени трех шимпанзе с NANBH давала результаты позитивной гибридизации для амплифицируемых последовательностей ожидаемого размера (161 и 586 нуклеотидов для 81 и 36 и 37b соответственно), в то время как контрольные шимпанзе давали негативные результаты гибридизации. Те же результаты были достигнуты при повторении этого эксперимента три раза.
Анализ нуклеиновых кислот и РНК из плазмы проводился в соответствии с вышеприведенной процедурой, используя праймеры и пробу из клона 81. Плазмы были из двух шимпанзе с NANBH, из контрольной шимпанзе и собранные плазмы из контрольных шимпанзе (объединенные). Обе NANBH плазмы содержали нуклеиновые кислоты/РНК, которые давали позитивные результаты в анализе PCR амплификацией, в то время как обе контрольные плазмы давали негативные результаты. Эти результаты последовательно получены несколько раз.
Дефектные вирусы, как известно, встречаются среди РНК вирусов. Используя технологию PCR, возможно сконструировать праймеры для амплификации последовательностей HCV генома. При анализе амплифицированных продуктов ожидают, что возможно идентифицировать дефектные версии вирусного генома так же, как и вирусные виды дикого типа. Соответственно, используя два праймера, основываясь на известной HCV последовательности, можно предсказать точно ожидаемый размер продукта PCR. Любые большие виды, наблюдаемые при электрофорезе в геле и анализе гибридизацией, могли бы представлять потенциальные вариантные геномы. Альтернативно, любой меньшего размера вид, наблюдаемый в подобной манере, может представлять дефектные агенты. Анализы этих типов были бы полезны в подтверждении точного происхождения известной HCV последовательности, является ли она действительно вирусной последовательностью дикого типа или дефектным геномом. Технологии и методы для этих анализов хорошо известны в науке, описаны ранее. Эта методология будет давать возможность тому, кто квалифицирован в этой области, получать родственные (дикого типа или дефектные) формы вирусного генома.
Детекция последовательностей в захватывающих частицах, которые, когда их амплифицировали при помощи PCR, гибридизовались с HCV кДНК, происходящей из клона 81
РНК в захватывающих частицах получена как описано ниже. Анализ последовательностей, которые гибридизуются с HCV кДНК, происходящей из клона 81, проводили, используя процедуру PCR амплификации как описано выше, за исключением того, что гибридизационная проба была меченый каназой олигонуклеотид, происходящий от клона 81 последовательности кДНК. Результаты показали, что амплифицированные последовательности гибридизуются с кДНК пробой.
Частицы захватывались из HCV инфицированной плазмы шимпанзе, используя полистереновые гранулы, покрытые иммуноочищенными антителами, направленными против полипептида, кодируемого
в клоне 5-1-1. Процедура получения препарата иммуноочищенных антител описана в EPO publication N 318216, которая в целом является собственностью настоящего представителя и которая вынесена здесь в ссылки. Вкратце, HCV полипептид, кодируемый клоном 5-1-1, экспрессировался как слитый полипетид с супероксид дисмутазой (SOD). Это осуществлялось субклонированием клона 5-1-1 кДНК инсерции в экспрессионный вектор pSODcf1 (Steimer и др. (1986)). ДНК, выделенную из pSODcf1, обрабатывали BamHI и EcoRI, и с линейной ДНК, образованной этими рестрикционными ферментами, лигировали следующий линкер:
5' GAT CCT GGA ATT CTG ATA AGA CCT TAA GAC TAT TTT AA 3'.
После клонирования выделяли плазмиду, содержащую инсерцию. Плазмиду, содержащую инсерцию, рестрицировали EcoRI. HCV кДНК инсерцию в клоне 5-1-1 вырезали EcoRI и лигировали в линеаризованную EcoRI плазмидную ДНК. Смесь ДНК использовали для трансформации штамма E.coli D1210 (Sadler и др., (1980)). Рекомбинанты с 5-1-1 кДНК в правильной ориентации для экспрессии ОРС определяли рестрикционным картированием и сиквенсом нуклеотидов. Рекомбинантные бактерии одного клона индуцировали для экспрессии SOD-NANB5-1-1 полипептида выращиванием бактерий в присутствии IPTG. Слитный полипетид очищали из рекомбинантной E.coli различной экстракцией клеточных экстрактов мочевиной, после чего следовала хроматография на анионно-катионной обменной колонке. Очищенный SOD-NANB5-1-1 полипептид прикрепляли к нитроцеллюлозной мембране. Антитела в образцах HCV инфицированных сывороток адсорбцировали с матрикссвязанным полипептидом. После отмывки, чтобы удалить неспецифически связавшиеся материалы и несвязавшиеся материалы, связанное антитело освобождали от связанного полипептида.
cPCR метод для детекции HCV РНК в печени и в сыворотке индивидуумов с NANBH
Достоверность и выгодность модифицированной формы PCR анализа, т.е. cPCR анализа, для детекции HCV инфекции определяли исполнением анализа на тотальной РНК печени и на сыворотке из инфицированных индивидуумов. В cPCR анализе предположительную вирусную РНК в образце обратно транскрибировали в кДНК обратной транскриптазой, сегмент полученной кДНК затем амплифицировали, используя модифицированную версию технологии, описанную Saiki и др. (1986). Праймеры для технологии происходили из HCV РНК, которую можно идентифицировать семейством HCV кДНК, приведенных здесь. Амплифицированный продукт, соответствующий HCV РНК, обнаруживали, используя пробу, берущую начало из семейства HCV кДНК, приведенного здесь.
cPCR/HCV анализ, использованный в этих исследованиях, выполняли используя следующие методы приготовления РНК, обратной транскрипции РНК в кДНК, амплификации специфических сегментов кДНК PCR и анализа продуктов PCR.
РНК экстрагировали из печени, используя гуанидин изотиоционатный метод получения тотальной РНК, описанный Maniatie и др. (1982).
Для того чтобы выделить тотальную РНК из плазмы, плазму разбавляли в пять - десять раз TENB (0,1 М NaCl, 50 мМ трис-HCl, pH 8,0, 1 мМ ЭДТА) и инкубировали с раствором протеиназа К/ДДС (0,5% ДДС, 1 мг/мл протеиназы K, 20 микрограмм/мл поли A носителя) в течение 60-90 минут при 37oC. Образцы экстрагировали один раз фенолом (pH 6,5), полученную органическую фазу реэкстрагировали один раз TENB, содержащим 0,1% ДДС, и водные фазы обеих экстракций объединяли и экстрагировали дважды равным объемом фенол/CHCl3/изомиловым спиртом (1: 1 (99:1)). Полученные водные фазы экстрагировали равным объемом CHCl3/изомиловый спирт (99:1) дважды и осаждали этанолом, используя 0,2 М ацетат натрия, pH 6,5 и 2,5 объема 100% этанола, осаждение в течение ночи при -20oC.
кДНК, использованную в качестве образца для реакции PCR, готовили, используя скопированные образцы для приготовления соответствующих кДНК. Каждый образец РНК, содержащий либо 2 микрограмма денатурированной нагреванием тотальной РНК из печени шимпанзе, РНК из 2 микролитров плазмы, либо 10% РНК экстрагированной из 10 мм х 4 мм цилиндрика биопсий печени человека, инкубировали в 25 микролитрах реакционной смеси, содержащей 1 микромоль каждого праймера, 2 миллимоль каждого дезоксирибонуклеотид трифосфата (дНТФ), 50 миллимоль Трис-HCl, pH 8,3, 5 миллимоль MgCl2, 5 миллимоль дитиотритол (ДТТ), 73 миллимоль KCl, 40 единиц ингибитора РНКаз (RNASIN) и 5 единиц обратной транскриптазы AMV. Инкубацию проводили 60 минут при 37oC. После синтеза кДНК реакционные смеси разводили 50 микролитрами деионизованной воды (DIW), кипятили в течение 10 минут и охлаждали во льду.
Амплификацию сегмента HCV кДНК выполняли используя два синтетических олигомерных 16-мерных праймера, чьи последовательности происходили из HCV кДНК клонов 36 (антисмысловой) и 37b (смысловой). Последовательность праймера из клона 36 была:
5' GCA TGT CAT GAT GTA T 3'.
Последовательность праймера из клона 37b была:
5' ACA ATA CGT GTG TCA C 3'.
Праймеры использовались в конечной концентрации 1 микромоль каждый. Для того чтобы амплифицировать сегмент кДНК HCV, которая фланкирована праймерами, образцы кДНК инкубировали с 0,1 микрограммами РНКазы A и PCR реагентами кита Perkin Elmer Cetus PCR (N 801-0043 или N 801-0055) в соответствии с инструкциями производителя. Реакцию PCR выполняли либо за 30 циклов, либо за 60 циклов в термальном циклизаторе Perkin Elmer Cetus DNA. Каждый цикл состоял из 1-мин. стадии денатурации при 94oC, стадии отжига 2 мин при 37oC, стадии элонгации 3 мин при 72oC. Однако стадия элонгации в окончательном цикле (30 или 60) была 7 мин, а не 3 мин. После амплификации образцы экстрагировали равным объемом фенол/хлороформ (1:1), после чего следовала экстракция равным объемом хлороформа и затем образцы осаждали этанолом, содержащим 0,2 М ацетат натрия.
Продукты cPCR анализировали следующим образом. Продукты подвергали электрофорезу в 1,8% щелочных агарозных гелях в соответствии с Murakawa и др. (1988) и переносили на Zetam Probe бумагу (Biokad Corp.) путем блоттинга гелей в течение ночи в 0,4 М NaOH. Блоты нейтрализовали в 2 х SSC (1 х SSC содержит 0,15 М NaCl, 0,015 М цитрат натрия), прегибридизовали в 0,3 М NaCl, 15 мМ натрий фосфатном буфере, pH 6,8, 15 мМ ЭДТА, 1,0% ДДС, 0,5% обезжиренным молоком (Cornation Co.) и 0,5 мг/мл разрушенной ультразвуком денатурированной ДНК из спермы лосося. Блоты, которые надо было анализировать на HCV кДНК фрагменты, гибридизовали с 32P-меченой пробой, образованной ник-трансляцией последовательности HCV кДНК инсерции в клоне 35, описанном EPO publication N 318216. После гибридизации блоты отмывали в 0,1 х SSC (1 х SSC содержит 0,15 М NaCl, 0,01 М Na цитрат) при 65oC, высушивали и авторадиографировали. Ожидаемый размер продукта 586 нуклеотидов длины, продукты, которые гибридизовались с пробой и мигрировали в гелях в масштабах этого размера считали позитивными на вирусную РНК.
Как контроль cPCR праймеры, предназначенные для амплификации альфа-1 антитриптозиновой мРНК, использовали для проверки присутствия РНК в каждом анализированном образце. Кодирующий район альфа-1 антитриптозинового гена описан у Rosenberg и др. (1984). Синтетические олигомерные 16-мерные праймеры, предназначенные для амплификации 365 нуклеотидного фрагмента кодирующего района альфа-1 антитриптозина, происходили от нуклеотидов 22-37 (смысловой) и нуклеотидов 372-387 (антисмысловой). Продукты PCR обнаруживали, используя 32P-ник-транслированную пробу, которая лежит между и не включает последовательностей с DNA/PCR праймеров.
Благодаря исключительной чувствительности реакции PCR все образцы прогоняли минимум три раза. Все ложные позитивные сигналы элиминировались при проведении следующих предосторожностей: 1) исключение попадания аэрозолей путем использования завинчивающихся крышек у пробирок с резиновыми O-формы прокладками-изоляторами, 2) раскапывание Ranin Micromemm пипетками со сбрасывателями со свободно передвигающимися поршнями/капиллярами и 3) выбор олигонуклеотидных последовательностей для кДНК и РНК праймеров из двух неприлежащих кДНК клонов.
Детекция HCV РНК в образцах печени методом cPCR
cPCR анализ выполняли на тотальной РНК, выделенной из печени трех шимпанзе, инфицированных агентом NANBH в условиях лаборатории, и из биопсий печени итальянских пациентов с диагнозом хронического NANBH.
Фиг. 24 показывает результаты cPCR анализа, используя 1 микрограмм каждого препарата тотальной РНК печени. РНК выделяли из образцов печени шимпанзе в хронической фазе инфекции (910) (строка 1), двух шимпанзе в острой фазе инфекции (1028 и 508) (строки 2 и 3 соответственно). PCR выполняли на образцах дорожек 1 - 3 в течение 30 циклов и авторадиограммы блотов, содержащих эти дорожки, экспозировали 5 часов. кДНК из 1 микрограмма тотальной РНК инфицированного животного в острой фазе (дорожка 4) 1028 и трех инфицированных шимпанзе (дорожки 5 - 7) амплифицировали в течение 60 циклов, и авторадиограммы, содержащие те дорожки, экспозировали 7 дней. 32P-меченая переваренная MspI pBR322 ДНК служила маркером на всех авторадиограммах. Можно видеть из этих результатов, что кДНК, соответствующая HCV РНК, видна только в образцах из шимпанзе с NANBH, острой ли, хронической ли формах (дорожки 1, 3 и 4). Продукты cPCR на этих дорожках мигрировали между маркерными фрагментами 527 и 622 нуклеотидов (не показано).
Фиг. 24B показывает результаты cPCR анализа, используя 10% РНК, экстрагированной из 10 мм х 4 мм цилиндриков биопсий печени из 15 пациентов с хроническим NANB (дорожки 1 - 15), одного пациента с криптогенным заболеванием печени (дорожка 16) и одного контрольного образца из пациента с хроническим гепатитом B (дорожка 17). Амплификацию PCR проводили за 30 циклов и авторадиограммы блотов экспозировали 4 дня, за исключением того, что дорожку 1 экспозировали 15 часов. Как видно из результатов, 9/15 (60%) человеческих образцов позитивно на HCV РНК (дорожки 1, 2, 4, 6, 7, 10-13). Один пациент с диагнозом криптогенной болезни печени (дорожка 16) и один пациент с хронической HBV инфекцией (дорожка 17) относительно негативны в cPCR анализе.
Сравнение HCV/cPCR анализа биопсий из печени человека и RIA из сыворотки, используя HCV C100-3 полипептид
SOD/HCV C100-3 полипептид (также названный C100) является рекомбинантным слитым полипетидом, который содержит 363 вирусных аминокислоты. Полипептид используется для детекции антител на HCV (см. Kuo и др., 1989). Метод приготовления C100 описан в EPO publication N 318216.
Радиоиммунный анализ, используя C100, выполнялся на сыворотке, собранной из тех же 17 пациентов, чьи образцы печени были подвергнуты HCV/cPCR анализу, как описано выше. Сыворотку отбирали в тот же день, что и биопсин печени. Анализ выполняли по существу как описано в EPO publication N 318216, которая полностью является собственностью уполномоченного и вынесена здесь в ссылки. Вкратце, микротитрационные плашки (иммулон 2 Removeawell strips) покрывали 0,1 микрограммом очищенного C100. Чашки инкубировали 1 час при 37oC с образцами сыворотки (100 микролитров разведения 1:100) или подходящими контролями. После инкубации несвязавшийся материал удаляли, плашки отмывали и комплексы человеческих антител-C100 детектировали инкубацией с 125I-меченым античеловеческим иммуноглобулином овцы. Радиактивность определяли в каждой лунке.
Результаты RIA показали, что шестьдесят семь процентов этих образцов были позитивными на анти-C100 антитела. Сыворотка из пациента с диагнозом криптогенной болезни печени была позивной на анти-C100 антитела, хотя уровни вирусной РНК были недетектируемыми в печени пациента в этом образце. Уровень корреляции между присутствием анти-C100 антител и HCV РНК был семьдесят процентов, два пациента, негативные на антитела RIA, имели значительные уровни HCV РНК в своей печени (данные не показаны).
Результаты указывают, что вирус часто присутствует в печени пациентов с циркулирующими анти-C100 антителами, и подтверждает утверждения, что присутствие анти-C100 антител точно отражает подверженность HCV. Более того, взятые вместе, эти результаты указывают, что HCV этого типа отвечает за NANBH по меньшей мере в 75% пациентов данного исследования и что основные штаммы HCV в Италии, по-видимому, близко родственны штаммам, превалирующим в США.
HCV/cPCR анализ сывороток: детекция вирусной РНК в шимпанзе с острой фазой инфекции
Временная взаимосвязь выявлением повреждения печени, присутствием HCV РНК и присутствием антиHCV-антител проводили в сыворотке из двух инфицированных в лаборатории шимпанзе с NANBH (поз. 771 и 910). Повреждения печени были определены по уровням аланиновой аминотрансферазы (ALT); присутствие HCV РНК определяли HCV с PCR анализом, описанным выше, антиHCV-антитела детектировали, используя C100 RIA.
HCV/cPCR анализ выполняли на РНК, экстрагированной из 1 микролитра плазмы шимпанзе. Сыворотки отбирали из шимпанзе 771 на 25, 32, 70 и 88 дни после инфекции, PCR выполняли за 30 циклов и авторадиограмму экспозировали в течение 18 дней. Сыворотки отбирали из шимпанзе 910 на 11, 23 и 67 дни после инфекции, PCR выполняли за 60 циклов и авторадиограмму экспозировали 5 дней.
Результаты анализов показаны на фиг. 25A для шимпанзе 771 и фиг. 25B для шимпанзе 910. Из сравнения фиг. 25A и 25B кажется, что ранний, хорошо различимый пик ALT величин во время острой фазы гепатита коррелирует с присутствием вирусной РНК в инфицированном индивидууме.
Эти данные также указывают, что присутствие РНК HCV, которая указывает состояние времени, предшествует присутствию антиHCV-антител. Шимпанзе 771 (фиг. 25A) демонстрирует рано выявленный острый эпизод при переливании крови с NANBH за 28 дней, как охарактеризовано первым пиком уровней ALT. HCV РНК обнаруживали в сыворотках, отобранных на 25 и 32 день. Однако во время этой острой фазы антиHCV-антитела отсутствовали. В противоположность этому на 70 день HCV РНК находилась ниже экспериментального уровня детекции и антиHCV-антитела возрастали. На 88 день HCV РНК оставалась недетектируемой, в то время как антиHCV-антитела значительно возрастали над антителами 70 дня.
Полученные из сыворотки шимпанзе 910 результаты были несколько подобны по Паттерну, хотя время индукции HCV антител при инфекции не детектировалось во время острой фазы болезни, которое увеличивалось по меньшей мере до 67 дней, антиHCV-антитела, детектируемые RIA на 11 день, были вызваны пассивной иммунизацией животного 910 антителами из плазмы, используемой для инокуляции животного. АнтиHCV-антитела были обнаружены в шимпанзе 910, в сыворотке во время поздней, хронической фазы инфекции (данные не показаны).
Следует заметить, что низкие значения ALT в плазме из индивидуумов с хроническими NANBH не обязательно коррелируют со слабой продукцией вируса. Пул 17 различных образцов плазмы, отобранных от шимпанзе 910 больше чем за период двух - трех с половиной лет после инокуляции, исследовался на уровни и HCV РНК. ALT значения образцов не превышали 45 мед./мл, тем не менее титрование указало на высокие титры HCV (3 х 106 СТД/мл). cPCR проводили за 30 циклов и авторадиограмму экспозировали 15 часов, PCR анализ ясно показал присутствие вирусной РНК (данные не показаны).
HCV/cPCR анализ сыворотки: детекция вирусной РНК в острой фазе инфекции у людей
Плазму из хирургических пациентов, собранную во время раннего острого NANBH, исследовали на HCV РНК и антиHCV-антитела, используя HCV/cPCR анализ с C100-RIA соответственно. HCV/cPCR анализ проводили используя 1 микролитр плазмы из пациента и из четырех контролей людей с известными родословными, cPCR выполняли за 30 циклов и после гибридизации и отмывания авторадиографию экспонировали восемь часов.
Результаты показали, что сыворотки, отобранные из хирургических пациентов во время острой инфекции, содержали высокий уровень вирусной РНК, и что антиHCV-антитела не детектировались C100-RIA (данные не показаны). Плазма при острой фазе из хирургических пациентов, как было известно, имеет высокий титр инфекционных агентов NANBH (106,5 CID/мл, как определено Feinstone и др. (1981), Feinstone и др., 1983). Следует заметить, однако, что этот пациент действительно был серо-скрытным по отношению к антиHCV-антителам С100-RIA приблизительно 9 месяцев после инфекции. Сыворотки из контрольных плазм пациентов с родословной были негативными как в HCV/cPCR анализе, так и C100-RIA.
Чувствительность HCV/cPCR анализа
Чувствительность HCV/cPCR анализа определяли анализом десятикратных серийных разведений пула плазмы известного титра. Плазма шимпанзе имела титр ~ 3 • 105 CID/мл, и РНК экстрагировали из десятикратных разведений 1 микролитра плазмы. cPCR выполняли за 30 циклов и после гибризации и отмывки авторадиограмму экспонировали 15 часов. Продукты cPCR, полученные при амплификации ~ 300, ~30 и ~3 CID HCV геномов показаны на дорожках 1 - 3 соответственно на фиг. 28. Образцы на дорожках 1 и 2 детектировали на авторадиограммах, экспонированных 2 часа.
Поскольку полный титр HCV в инфицированных индивидуумах как считают, приблизительно между 100 и 10000 CID/мл плазмы, эти данные предлагают, что HCV/cPCR анализ, возможно, клинически полезен.
HCV/cPCR анализ вариантных HCV штаммов
Праймеры, состоящие из набора олигомерных 44-меров и набора олигомерных 45-меров, были сконструированы, чтобы амплифицировать штаммы HCV, подобные или идентичные изоляту HCV, из которого кДНК последовательность на фиг. 18 происходит. Лежащая в основе создания этих праймеров предпосылка - наше открытие того, что HCV является флавиподобным вирусом. Члены семейства Feaviviridae по сравнению с HCV имеют два главных консервативных набора аминокислотных последовательностей. TATPPG и QRRGR, в предположительном NS3 районе этих вирусов. Несколько другие меньшие наборы можно видеть, например, в случае GDD в предположительном районе NS5. Другие наборы определяли сравнением известных аминокислотных последовательностей с последовательностями HCV. Эту информацию вывели из последовательностей для нескольких членов Flaviviridae, которые были описаны, включая вирус японского энцефалита (Sumiyoshi и др. 1987), вирус желтой лихорадки (Rice и др., 1985), вирус денге типа 2 (Hahn и др., 1988), вирус денге типа 4 (Mackow, 1987) и вирус западного Нила (Castle и др., 1986). Консервативные аминокислотные последовательности и частота использования кодонов представлены в таблице 6 (последовательность праймера выбрана так, чтобы свести к минимуму количество вырожденных нуклеотидов на 3-конце последовательности праймера и максимально увеличить количество нуклеотидов на 3'-конце каждого праймера, которые точно совпадают с любой из возможных нуклеотидных последовательностей или их комплементов, кодирующих консервативные аминокислоты).
44-мерные и 45-мерные олигонуклеотидные праймеры сделаны так, что последовательности, кодирующие эти аминокислоты, были включены внутри праймера. Более того, они содержат вырожденность на 3'-конце каждого праймера и происходят из двух разных районов генома, которые присутствуют в клоне 406 (см. фиг. 27) и которые происходят из района, кодирующего предположительно NS3 HCV. Формула для олигонуклеотидных праймеров в этих наборах:
5' GAC TGC GGG GGC GAG ACT GGT TGT GCT CGC ACC GCC ACC CCX CC 3',
где X это A, T, G или G; и
5' TCT GTA GAT GCC TGG CTT CCC CCT GCC AGT CCT GCC CCG AGT YTG 3',
где Y это T или C.
HCV/cPCR анализ проводили используя эти праймеры, чтобы амплифицировать HCV РНК шимпанзе 910, плазмы. Метод этого анализа по существу проводился как описано в разделе выше, за исключением того, что 44- и 45-мерные наборы олигомерных праймеров заменили на праймеры, происходящие из клонов 36 и 37b. В дополнение, детекцию амплифицированной HCV кДНК проводили гибридизацией с пробой, происходящей из клона 40a, последовательность которого показана на фиг. 29.
Пробу готовили амплификацией сегмента клона 40a, используя метод PCR, описанный выше, и 18-мерные праймеры, содержащие следующие последовательности:
5' GAG ACA TCT CAT CTT CTG 3'
и
5' GAG CGT GAT TGT CTC AAT 3'.
После амплификации препарата пробы метили 32P-ник-трансляцией.
Фиг. 30 показывает авторадиографии Саузерн блотов, гибридизованных с последовательностью, происходящей из клона 40a. 32P-меченые MspI расщепленная ДНК фрагменты pBR 322 служили в качестве маркеров (дорожка 1). Предсказанный размер продукта PCR, полученного амплификацией, используя эти праймеры, - 490 нуклеотидов (пт). Повторные реакции показаны на дорожках 2 и 3.
Анализ вариантов 5'-района HCV
На основании модели флавивирусов, 5'-район HCV кДНК который фланкирован районами, представленными в клонах ag30a и K9-1, кодирует сегмент предположительно белка(ов) оболочки и/или матрикса. (E/M). Сыворотки, полученные из шимпанзе, из которых получали HCV кДНК "с" библиотеку, были сконструированы таким образом, чтобы быть анализированными HCV/cPCR для определения, присутствуют ли внутри этого района мишени варианты.
HCV/cPCR анализ выполняли по существу как описано выше, для выделения клона 5'-32, за исключением использованных праймеров и проб. Фиг. 32 показывает взаимосвязь праймеров и проб (и клонов, из которых они произошли) с праймерами и пробами района мишени HCV кДНК. Один набор PCR праймеров, ag30a16A и K91Env16B, происходил из клонов ag30a и K9-1, которые лежат выше и ниже соответственно последовательности мишени. Ожидаемая длина продукта PCR, праймированного аg30a16A и K91Env16B, 1,145 тпн, основываясь на подтвержденной последовательности HCV кДНК. Два других набора праймеров PCR, покрывающих район, амплифицируемый, используя аg30a16A и K91Env16B, и перекрывающих каждый другой, также использовали для амплификации HCV РНК сыворотки. Таким образом, в этом случае реакции PCR проводились, используя как один набор праймеров аg30a16A и CA156e16B, так и второй набор праймеров CA156e16A и 91Env16B. Размеры ожидаемого продукта PCR для этих пар были 615 нуклеотидов (нт) и 683 нт соответственно. Cписки праймеров, клоны, из которых они происходят и последовательности праймеров приведены в таблице 7.
Пробы для всех трех HCV/cPCR продуктов состояли из 32P-меченых частей HCV кДНК, которые были приготовлены амплификацией PCR района клона 216 (используя CA216a16A и 216a16B как праймеры) и клона 84 (используя CA84a16A и CA84a16B как праймеры), 32P включали в продукты PCR ник-трансляцией. Эти пробы не перекрывались с праймерами, используемыми в реакциях HCV/cPCR.
Фиг. 33 показывает авторадиографию Саузерн блота, в котором HCV/cPCR продукты гибридизованы с 32P-мечеными пробами. HCV/cPCR продукты, наращиваемые с праймеров аg30a16A и K91Env16 (дорожка 1) были приблизительно 1,1 тпн, не наблюдалось никаких других продуктов PCR при 15-часовой экспозиции. Продукты, наращиваемые с наборов праймеров аg30a15A (CA156e16B) (дорожка 2) и CA156e16a/KEnv16B (дорожка 3), были приблизительно 625 нт и 700 нт соответственно. Размер продуктов PCR определяли сравнением с относительной миграцией фрагментов, полученных при переваривании PBR 22 MspI и PhiX 174, расщепленной HalIII (дорожка 5).
Вышепроведенное исследование обнаруживает инсерции или делеции вплоть до приблизительно 20 нт -50 нт и перестройки ДНК, изменяющие размер ДНК мишени. Результаты на фиг. 33 подтверждают, что есть только один основной вид кДНК, происходящий из E/M района HCV в сыворотках шимпанзе.
Амплификация с целью клонирования последовательностей HCV кДНК, используя PCR и праймеры, происходящие из консервативных районов геномных последовательностей флавивирусов
Наше открытие того, что HCV является флавиподобным вирусом, позволило использовать стратегию клонирования неохарактеризованных HCV кДНК последовательностей, используя PCR технологию и праймеры, происходящие из районов, кодирующих консервативные аминокислотные последовательности флавивирусов. В целом один из праймеров происходит из определенной HCV геномной последовательности, а другой праймер, который фланкирует район несеквенированного HCV полинуклеотида, происходит из консервативного района генома флавивирусов. Геномы флавивирусов, как известно, содержат консервативные последовательности внутри NS1 и E полипептидов, которые кодируются в 5'-районе генома флавивирусов. Таким образом, чтобы изолировать кДНК последовательности, происходящие от предположительно сравнимых районов HCV генома, вышерасположенный праймер сконструирован такой, чтобы он происходил из консервативных последовательностей внутри этих полипептидов флавивирусов. Нижерасположенный праймер происходит от вышерасположенного конца известной части HCV кДНК.
Из-за вырожденности кода возможны замены у флавивирусных проб по сравнению с соответствующей HCV геномной последовательностью. Поэтому используют стратегию, подобную описанной Lee (1988). Процедура по Lee использует смешанные олигонуклеотидные праймеры, комплементарные продуктам обратной трансляции аминокислотной последовательности, последовательности смешанных праймеров принимают во внимание каждый вырожденный кодон в консервативной аминокислотной последовательности.
Три набора смесей праймеров составлены основываясь на аминокислотных гомологиях, найденных в нескольких флавивирусах, включая денге-2,4 (D-2,4), вирус японского энцефалита (JEV), желтой лихорадки (YF) и западного Нила (WN). Смесь праймеров, происходящая из наиболее вышерасположенной консервативной последовательности (5'-I), основана на аминокислотной последовательности gly-trp-gly, которая является частью консервативной последовательности asp-arg-gly-trp-gly-aspN, обнаруженной в E белке Д-2, JEV, YF и WN. Следующая смесь праймеров (5'-2) основана на правее расположенной последовательности в E белке phe-asp-gly-asp-ser-tyr-ileu-phe- gly-asp-ser-tyr-ilen и происходит от phe-gly-asp, эта консервативная последовательность присутствует в D2, JEV, YF и WN. Третья смесь праймеров (5'-3) основана на аминокислотной последовательности arg-ser-cys, которая является частью консервативной последовательности cys-cys-arg-ser-cys в NS 1 белке Д-2, Д-4, JEV, YF и WN. Индивидуальные праймеры, которые образуют смесь 5'-3, показаны на фиг. 45. В дополнение к варьирующим последовательностям, происходящим из консервативного района, каждый праймер в каждой смеси также содержит константный район на 5'-конце, который содержит последовательность, кодирующую сайты для ферментов рестрикции HindIII MboI и EcoRI.
Правее расположенный праймер ssc5h20A происходит из нуклеотидной последовательности клона 5h, который содержит кДНК с последовательностями, которые перекрывают последовательности клонов 14i и 11b. Последовательность ssc5h20A:
5' GTA ATA TGG TGA CAG AGT CA 3'.
Можно также использовать альтернативный праймер, ssc5h34A. Этот праймер происходит из последовательности клона 5h и дополнительно содержит нуклеотиды на 5'-конце, которые создают рестрикционный сайт, облегчая таким образом клонирование. Последовательность ssc5h34A:
5' GAT CTC TAG AGA AAT CAA TAT GGT GAC AGA GTC A 3'.
Реакция PCR, которая первоначально описана Saiki и др., 1986, проводится по существу как описано Lee и др., 1988, за исключением того, что образец для кДНК в РНК изолирован из печени инфицированной HCV шимпанзе или из вирусных частиц, выделенных из сыворотки HCV инфицированной шимпанзе. В дополнение условия отжига менее жесткие на первом раунде амплификации (0,6 М NaCl и 25oC), поскольку часть праймера, отжигающаяся с HCV последовательностью, только 9 нуклеотидов, и могут быть замены. Более того, если используют ssc5h34A, дополнительные последовательности, не происходящие из генома, имеют тенденцию дестабилизовать гибрид праймер - матрица. После первого раунда амплификации условия отжига могут быть более строгие (0,066 М NaCl и 32-37oC), поскольку амплифицированные последовательности теперь содержат районы, комплементарные или дупликаты праймеров. В дополнение первые 10 циклов амплификации проводят с фрагментом Кленова 1 при подходящих условиях для этого фермента. После завершения этих циклов образцы экстрагируют и проводят реакцию с Tag-полимеразой в соответствии с указаниями кита, как установлено Cetus/Perkin - Elmer.
После амплификации амплифицированные HCV кДНК последовательности детектируют гибридизацией, используя пробу, происходящую из клона 5h. Эта проба происходит из последовательностей, лежащих выше последовательностей, использованных в праймере, и не перекрываются с последовательностями из клона 5h, происходящих праймеров. Последовательность этой пробы
5' CCC AGC GGC GTA CGC GCT GGA CAC GGA GGT GGC CGC GTC GTC TGG CGG TGT TGT TCT CGT CGG GTT GAT GGC GC 3'.
Промышленное применение
Описанный здесь метод так же, как олигомеры, как пробы, так и праймеры, происходящие из HCV кДНК, и киты, содержащие их, полезны для точного, относительно простого и экономичного определения присутствия HCV в биологических образцах, особенно в крови, используемой для гемотрансфузии и в индивидуумах, подозрительных на HCV инфекцию. Более того, эти методы и олигомеры могут быть полезными для обнаружения на более ранних стадиях HCV инфекции, чем иммунологическими анализами, основанными на использовании рекомбинантных HCV полипептидов. Также гибридизационный анализ амплифицированного полинуклеотида обнаруживает HCV РНК в случайных образцах, которые являются антиHCV негативными. Таким образом, описанные здесь пробы и праймеры можно использовать амплифицированными в гибридизационных анализах в сочетании с иммуноанализами, основанными на HCV полипептидах, для более совершенной идентификации инфекций, обусловленных HCV, и HCV-инфицированных образцов, включая кровь.
Приведенная здесь информация позволяет иметь праймеры и/или пробы, которые берут начало из консервативных районов генома HCV. Обеспечение этими праймерами и пробами делает возможным основной метод, которым детектируются вариантные HCV штаммы и который будет использоваться при скрининге крови и ее продуктов.
Если использованные в методе праймеры происходят из консервативных районов генома, метод следует нацелить на детекцию и/или идентификацию вариантных штаммов HCV. Это, в свою очередь, должно вести к получению дополнительных иммунологических реагентов для детекции и диагноза HCV так же, как и к созданию полинуклеотидных реагентов для детекции и/или обработки HCV.
В дополнение к этому, наборы праймеров и проб, полученных из районов консервативных аминокислотных последовательностей флавивирусов и HCV, позволяют использовать универсальный метод детекции для этих инфекционных агентов.
Нижеследующие перечисленные материалы (приведенные в конце описания) сделаны на депозит в терминах Budapest Treaty with the American Type Culture Collection (АТСС), 12301 Parklawn Dr. Rockiville Maryland 20852 и названы следующими Accession номерами.










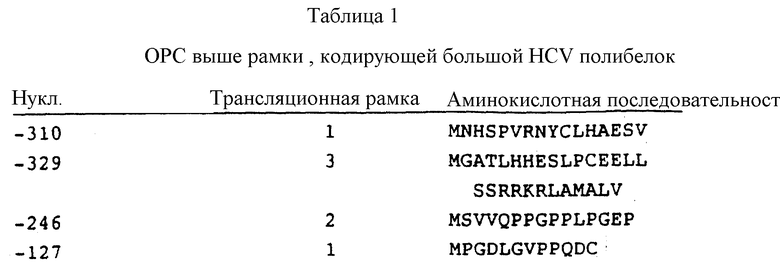
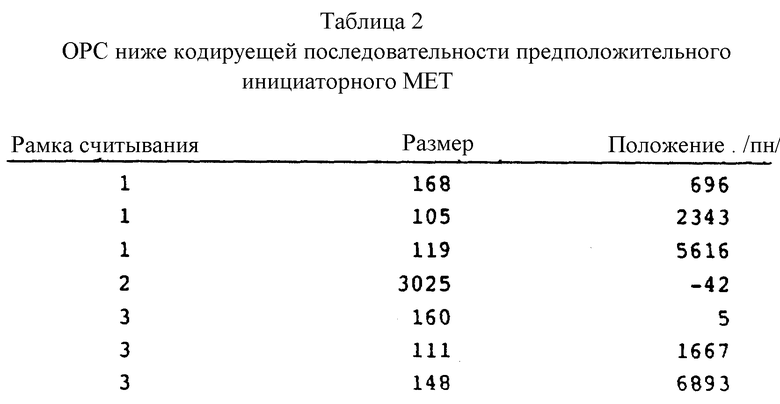
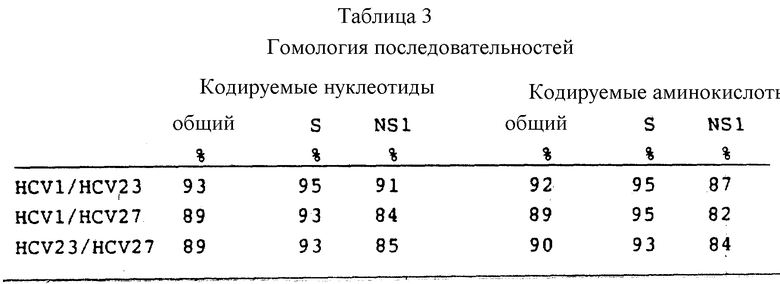



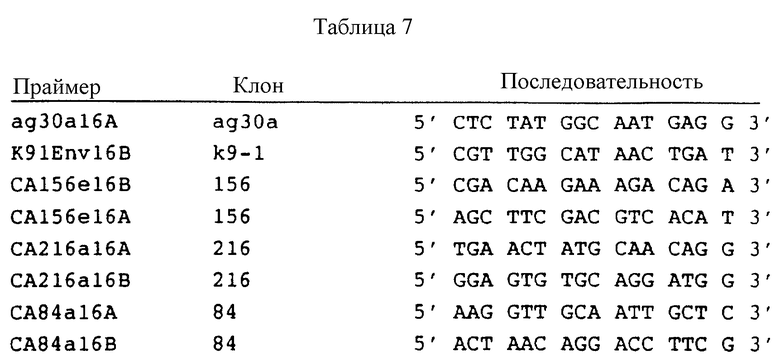
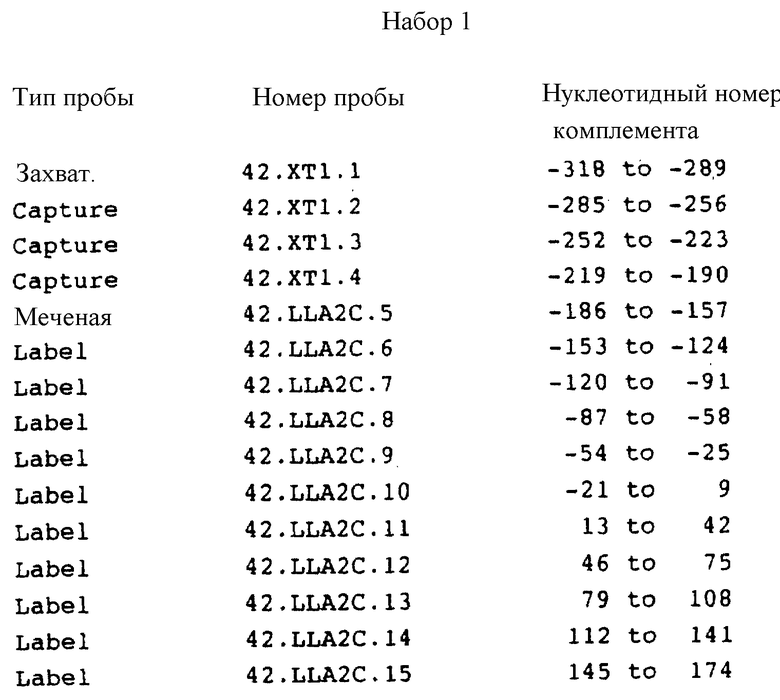
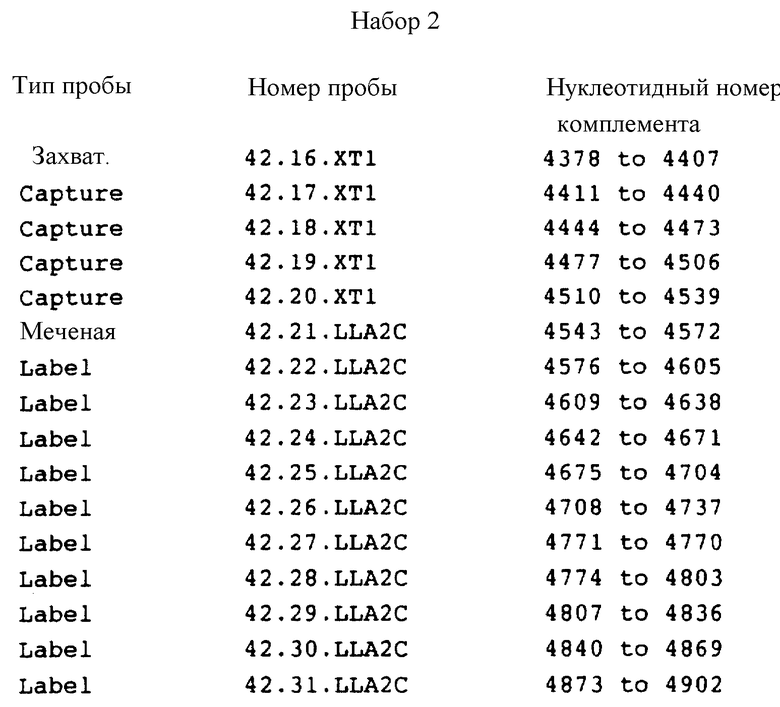

























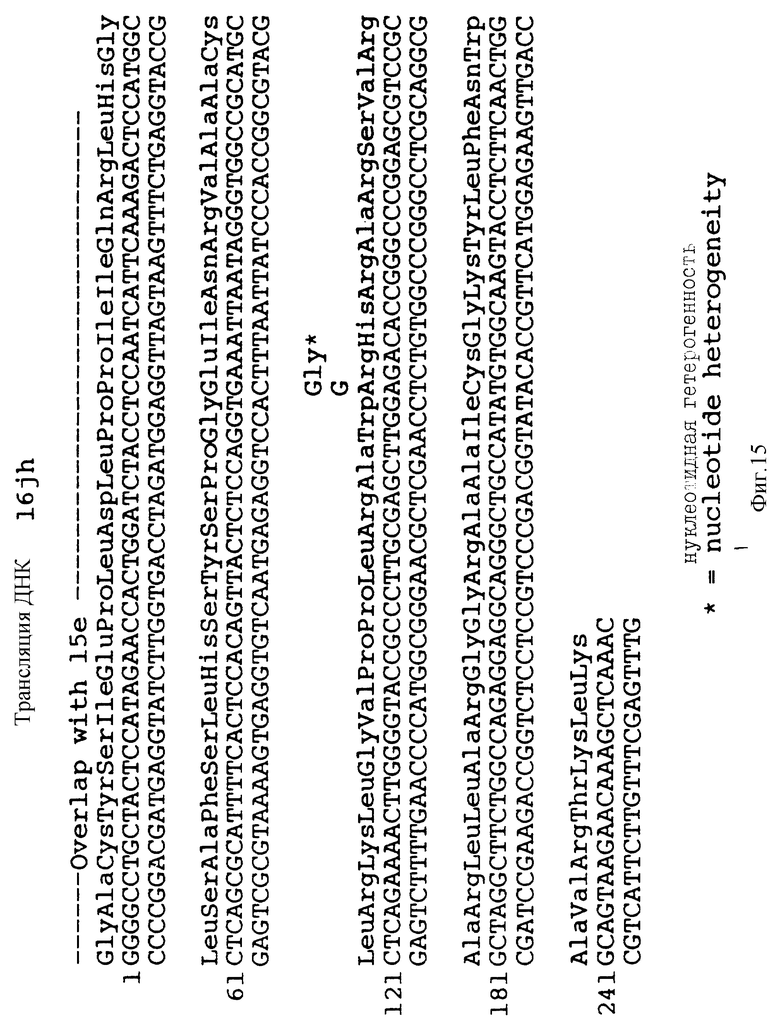






























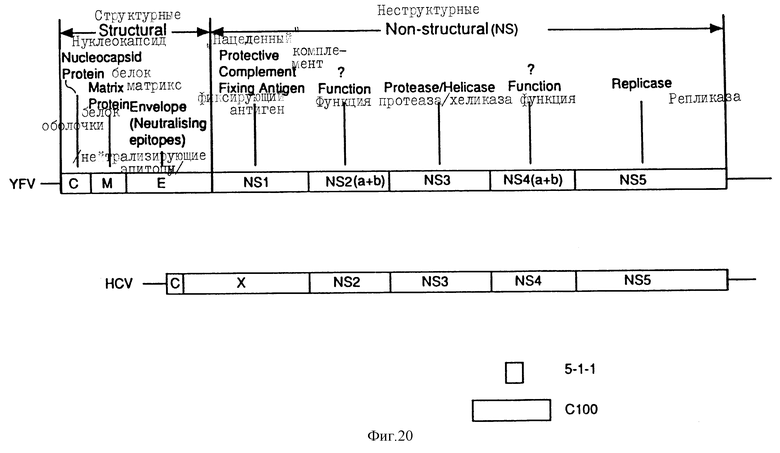



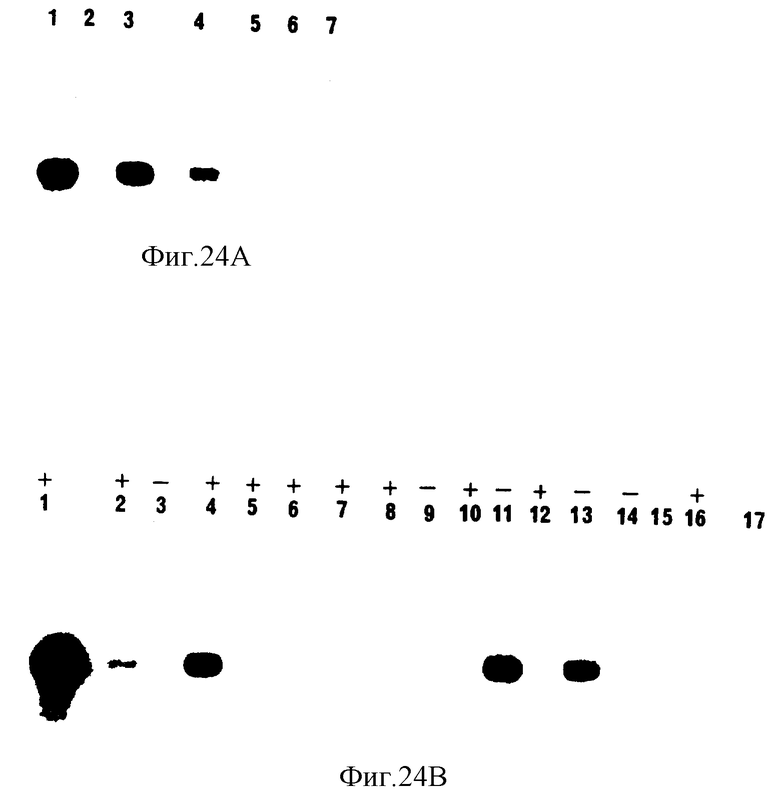
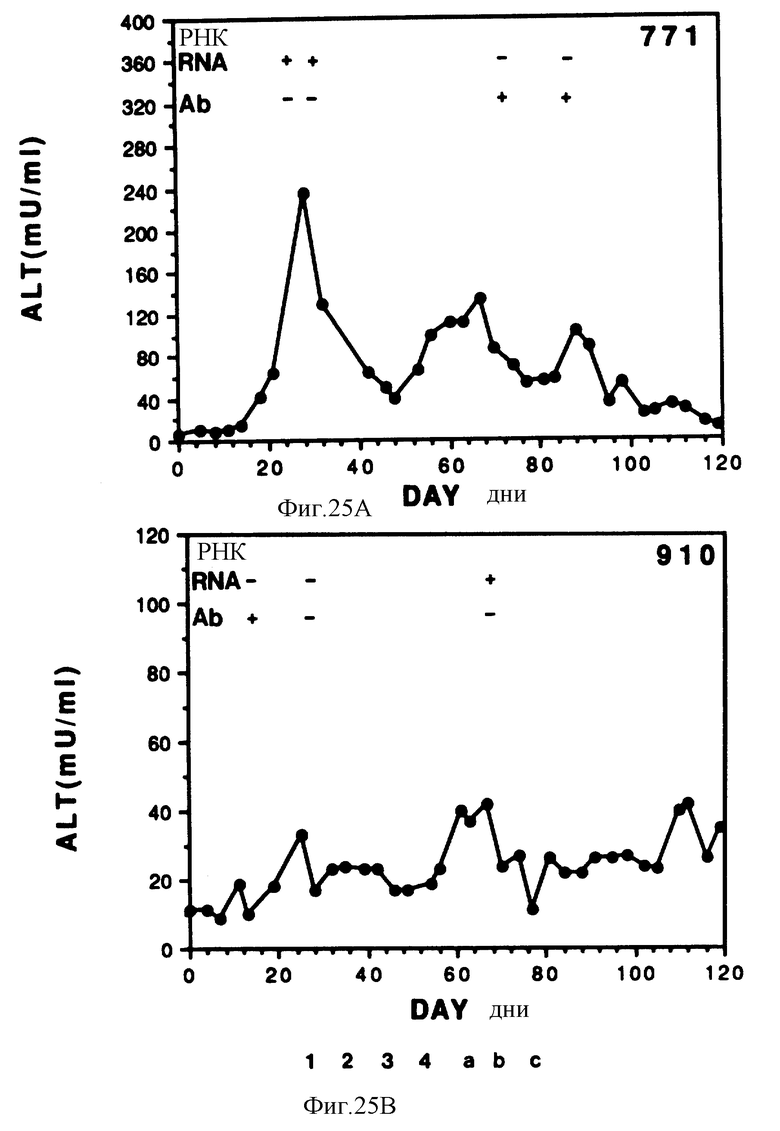







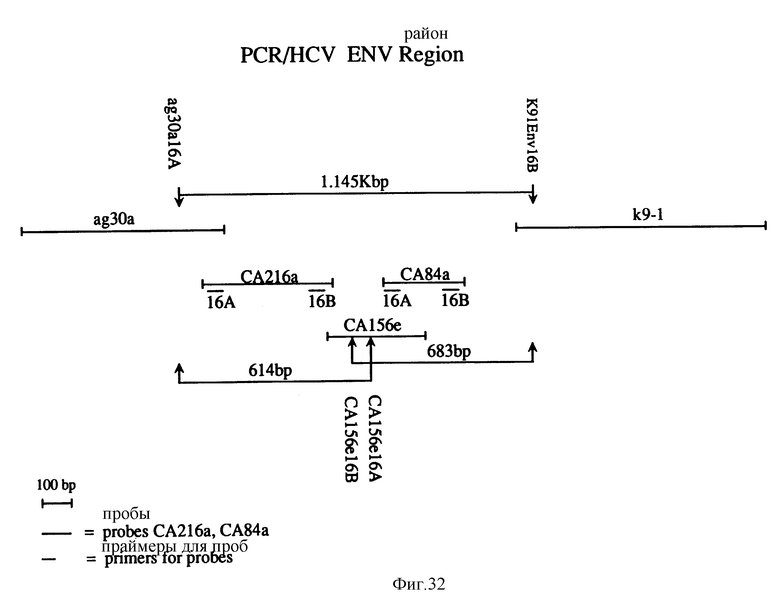
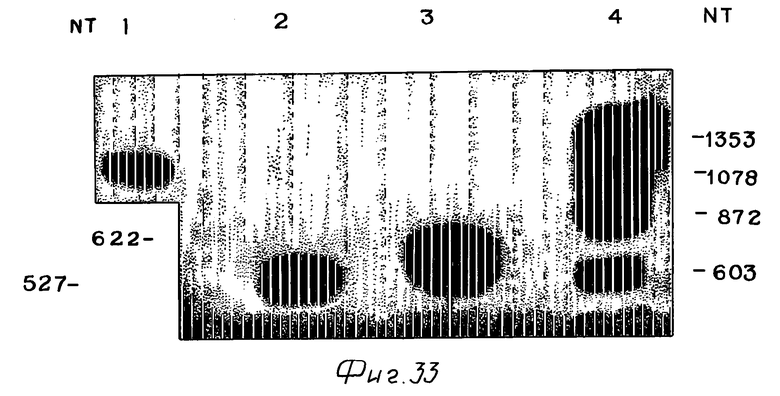





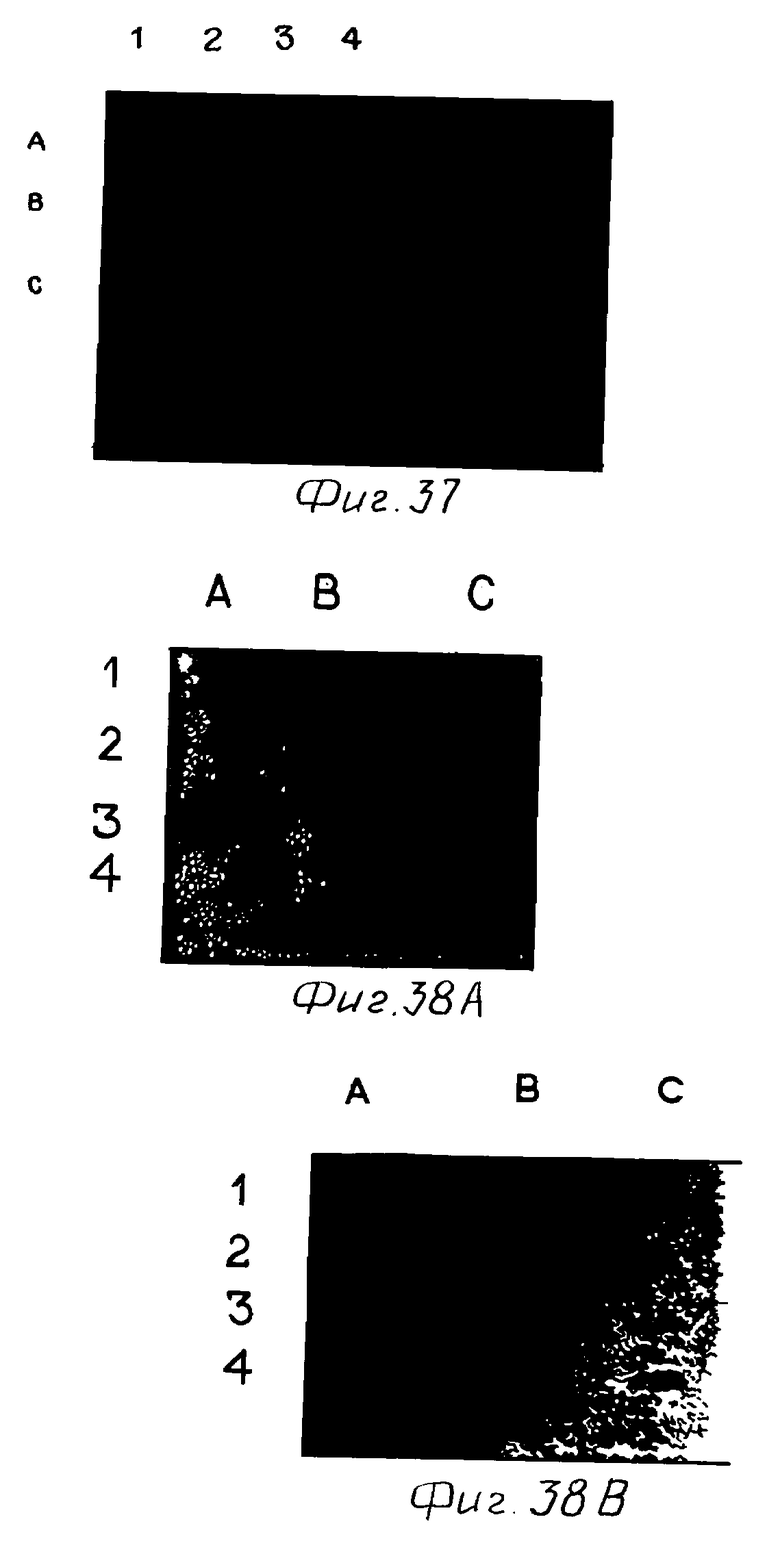


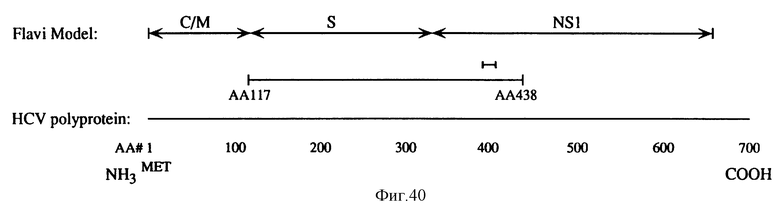







Изобретение относится к генной инженерии. Полинуклеотид, используемый для обнаружения вируса гепатита HCV, и его фрагменты показаны на фиг. 18. Праймер для синтеза ДНК, меченый зонд, несущий обнаруживаемую метку, и зонд захвата представляют собой фрагмент полинуклеотида. Проводят инкубирование полинуклеотидного фрагмента с образцом в условиях, обеспечивающих образование гибридизованных дуплексов между полинуклеотидным реагентом и нуклеиновой кислотой HCV. Обнаруживают образуемые дуплексы, перед инкубацией полинуклеотидного реагента с образцом нуклеиновую кислоту в образце могут дополнительно амплифицировать методом полимеразной цепной реакции с использованием праймера. Из образца крови, подозреваемого на содержание нуклеиновой кислоты HCV, получают нуклеиновую кислоту. Ее инкубируют с фрагментом полинуклеотида в качестве полинуклеотидного реагента в условиях, допускающих образование гибридизованных дуплексов между нуклеиновой кислотой HCV и указанным полинуклеотидным реагентом. Изобретение позволяет обнаруживать вирус гепатита HCV в биологических образцах. 15 с. и 30 з.п.ф-лы, 7 табл., 45 ил.
16 Фрагмент полинуклеотида по п. 10, отличающийся тем, что содержит нуклеотиды от положения 8920 до 8930, как показано на фиг.18.
Приоритет по пунктам:
18.05.89 по пп. 42 - 45;
04.04.90 по пп. 1 - 41.
| US 4683195 A (28.07.87) | |||
| US 4683202 A (28.07.87) | |||
| Картофелеуборочная машина | 1946 |
|
SU68465A1 |
| Способ определения глубин опускания зонда | 1950 |
|
SU92249A1 |
| ДАТЧИК ФАЗЫ | 0 |
|
SU388232A1 |
| ЭЛЕКТРИЧЕСКОЕ УСТРОЙСТВО | 0 |
|
SU293274A1 |
| Способ дифференциальной индикации вирусспецифических нуклеотидных последовательностей вируса герпеса простого типа 1 и 2. | 1989 |
|
SU1645302A1 |
| US 4341761 A, 1988 | |||
| US 4868105 A, 1989. | |||
Авторы
Даты
2000-02-20—Публикация
1990-05-18—Подача