Настоящее изобретение относится к выделению и идентификации новых генов пути биосинтеза спирамицинов и к новым полипептидам, участвующим в этом биосинтезе. Оно также относится к применению этих генов с целью увеличения степени продуцирования и чистоты получаемого спирамицина.
Изобретение относится также к применению этих генов для конструкции мутантов, которые могут приводить к синтезу новых антибиотиков или к производным формам спирамицинов. Изобретение относится также к молекулам, продуцируемым за счет экспрессии этих генов, и, наконец, к фармакологически активным композициям, включающим молекулу, продуцируемую за счет экспрессии таких генов.
Streptomyces представляют собой грамположительные филаментозные бактерии почвы. Они играют важную роль в деструкции и минерализации органических материалов благодаря большому разнообразию расщепляющих ферментов, которые они выделяют. Они обладают явлениями единственных в своем роде морфологических дифференцировок в прокариотах, сопровождающихся метаболической дифференцировкой, характеризующейся продуцированием вторичных метаболитов, обладающих огромным разнообразием химических структур и биологических активностей. Среди этих метаболитов находят спирамицины, продуцируемые в нативном состоянии бактерией Streptomyces ambofaciens.
Спирамицин представляет собой антибиотик семейства макролидов, пригодный как в ветеринарии, так и в медицине. Макролиды характеризуются наличием лактонового цикла, к которому привиты один или несколько сахаров. Streptomyces ambofaciens в нативном состоянии продуцирует спирамицины I, II и III (см. фиг.1), однако, антибиотическая активность спирамицина I отчетливо выше таковой спирамицинов II и III (Liu и др., 1999). Молекула спирамицина I образована лактоновым макроциклом, называемым платенолидом, и тремя сахарами: форозамином, микаминозой и микарозой (см. фиг.1). Антибиотическая активность спирамицинов возникает вследствие ингибирования протеинового синтеза в прокариотах за счет механизма, приводящего к связыванию антибиотика с бактериальной рибосомой.
Некоторые соединения, являющиеся членами семейства макролидов и имеющие также лактоновый цикл, можно использовать в области антибиотиков. Так, продукт FK506 обладает иммуносупрессорными эффектами и открывает перспективы терапевтического использования в области трансплантации органов, ревматоидного артрита и, вообще, в случае патологий, связанных с аутоиммунными реакциями. Другие макролиды, как авермектин, обладают инсектицидными и антигельминтными активностями.
Многочисленные пути биосинтеза, касающиеся антибиотиков, принадлежащих к различным классам, а также других вторичных метаболитов (согласно обзору, Chater K., 1990), на сегодняшний день уже изучены в случае Streptomyces. Однако знание путей биосинтеза спирамицинов в настоящее время является только очень частичным.
Биосинтез спирамицинов представляет собой сложный процесс, включающий многочисленные стадии и опосредуемый многочисленными ферментами (Omura и др., 1979а; Omura и др., 1979b). Спирамицины относятся к большому классу поликетидов, который объединяет комплексные молекулы, особенно изобилующие в микроорганизмах почвы. Эти молекулы объединяют не по аналогии структуры, а по некоторому подобию на уровне стадий пути их биосинтеза. В самом деле, поликетиды продуцируются комплексным рядом реакций, но в пути их биосинтеза совместно обладают рядом реакций, катализируемых ферментом или ферментами, называемыми “поликетидсинтазами” (PKS). В случае Streptomyces ambofaciens биосинтез лактонового макроцикла спирамицинов (платенолид) протекает через ряд из восьми модулей, кодируемых пятью разными генами PKS (Kuhstoss S., 1996; патент США 5945320). Спирамицины образуются из этого лактонового цикла. Однако различные стадии и ферменты, участвующие в синтезе сахаров, а также их связывание с лактоновым циклом и модификация этого цикла для получения спирамицинов на сегодняшний день остаются еще неизвестными.
В патенте США 5514544 описывается клонирование последовательности, называемой srmR, в Streptomyces ambofaciens. В этом патенте высказывается гипотеза, что ген srmR кодирует протеин-регулятор транскрипции генов, участвующих в биосинтезе макролидов.
В 1987 г. Richardson и сотрудники (Richardson и др, 1987) показали, что резистентность к спирамицину S. ambofaciens придается по меньшей мере тремя генами, причем эти последние были названы srmA, srmB и srmC. В патенте США 4886757 в особенности описывается характеристика фрагмента ДНК S. ambofaciens, содержащего ген srmC. Однако последовательность этого гена не была представлена. В 1990 г. Richardson и сотрудники (Richardson и др., 1990) предложили гипотезу о существовании трех генов биосинтеза спирамицина, близких к srmB. В патенте США 5098837 сообщается о клонировании пяти генов, потенциально участвующих в биосинтезе спирамицина. Эти гены были названы srmD, srmE, srmF, srmG и srmH.
Одна из больших трудностей при получении таких соединений, как спирамицины, заключается в том, что для получения относительно незначительного количества продукта необходимы очень большие объемы при ферментации. Следовательно, желательно иметь возможность повышать эффективность получения таких молекул в целях снижения издержек производства.
Путь биосинтеза спирамицинов представляет собой сложный процесс и желательно идентифицировать и ликвидировать паразитические реакции, которые могут протекать во время этого процесса. Целью такой манипуляции является получение более чистого антибиотика и/или повышение производительности. По этому поводу, Streptomyces ambofaciens в нативном состоянии продуцирует спирамицины I, II и III (см. фиг.1), однако, антибиотическая активность спирамицина I отчетливо выше таковой спирамицинов II и III (Liu и др., 1999). Следовательно, желательно иметь возможность располагать штаммами, продуцирующими только спирамицин I.
Ввиду коммерческого интереса к макролидным антибиотикам проводятся интенсивные исследования в отношении получения новых производных, в частности, аналогов спиромицинов, обладающих предпочтительными свойствами. Желательно иметь возможность генерировать в достаточном количестве медиаторы пути биосинтеза спирамицинов или производных спирамицинов, в частности, с целью получения гибридных антибиотиков, происходящих от спирамицинов.
Общее описание изобретения
Настоящее изобретение основано на клонировании генов, продукт которого участвует в биосинтезе спирамицинов. Изобретение относится, прежде всего, к новым генам пути биосинтеза спирамицинов и новым полипептидам, участвующим в этом биосинтезе.
Были клонированы гены пути биосинтеза и ассоциированные кодирующие последовательности и была определена последовательность ДНК каждого. Клонированные кодирующие последовательности в дальнейшем обозначают как orf1*c, orf2*c, orf3*c, orf4*c, orf5*, orf6*, orf7*c, orf8*, orf9*, orf10*, orf1, orf2, orf3, orf4, orf5, orf6, orf7, orf8, orf9c, orf10, orf11c, orf12, orf13c, orf14, orf15c, orf16, orf17, orf18, orf19, orf20, orf21c, orf22c, orf23c, orf24c, orf25c, orf26, orf27, orf28c, orf29, orf30c, orf31, orf32c, orf33 и orf34c. Функция протеинов, кодируемых этими последовательностями, в пути биосинтеза спирамицинов раскрывается в нижеприводимом обсуждении, которое поясняется фиг.4, 5, 6 и 8.
1) Первый объект изобретения относится к полинуклеотиду, кодирующему полипептид, участвующий в биосинтезе спирамицинов, отличающемуся тем, что последовательность вышеуказанного полинуклеотида представляет собой:
(а) одну из последовательностей SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 107, 109, 111, 113, 115, 118, 120, 141, 143, 145, 147 и 149,
(b) одну из последовательностей, образованных вариантами последовательностей (а);
(с) одну из последовательностей, происходящих от последовательностей (а) и (b) в соответствии с вырожденностью генетического кода.
2) Объектом настоящего изобретения также является полинуклеотид, гибридизующийся в условиях гибридизации высокой жесткости по меньшей мере с одним из полинуклеотидов согласно вышеприведенному п.1).
3) Изобретение относится также к полинуклеотиду, обладающему по меньшей мере 70%, 80%, 85%, 90%, 95% или 98% идентичности по нуклеотидам с полинуклеотидом, включающим по меньшей мере 10, 12, 15, 18, 20-25, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 350, 400, 450, 500, 550, 600, 650, 700, 750, 800, 850, 900, 950, 1000, 1050, 1100, 1150, 1200, 1250, 1300, 1350, 1400, 1450, 1500, 1550, 1600, 1650, 1700, 1750, 1800, 1850 или 1900 последовательных нуклеотидов полинуклеотида согласно вышеприведенному п.1).
4) Изобретение также относится к полинуклеотиду согласно вышеприведенному п.2) или 3), отличающемуся тем, что он выделен из бактерии рода Streptomyces.
5) Изобретение относится также к полинуклеотиду по вышеприведенному п.2), 3 или 4, отличающемуся тем, что он кодирует протеин, участвующий в биосинтезе макролида.
6) Изобретение также относится к полинуклеотиду по вышеприведенному п.2), 3) или 4), отличающемуся тем, что он кодирует протеин, обладающий активностью, подобной протеину, кодируемому полинуклеотидом, с которым он гибридизуется или обладает идентичностью.
7) Изобретение также относится к полипептиду, образующемуся в результате экспрессии полинуклеотида по вышеприведенному п.1), 2), 3), 4), 5) или 6).
8) Следующий аспект изобретения относится к полипептиду, участвующему в биосинтезе спирамицинов, отличающемуся тем, что последовательность вышеуказанного полипептида представляет собой:
(а) одну из последовательностей SEQ ID № 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 27, 29, 31, 32, 33, 35, 37, 38, 39, 41, 42, 44, 46, 48, 50, 51, 52, 54, 55, 56, 57, 58, 59, 61, 63, 65, 67, 69, 71, 73, 75, 77, 79, 81, 83, 85, 108, 110, 112, 114, 116, 117, 119, 121, 142, 144, 146, 148 и 150,
(b) одну из последовательностей, таких как указанные в п.(а), если не считать того, что по всей длине вышеуказанной последовательности одна или несколько аминокислот заменены, встроены или подвергнуты делеции без затрагивания функциональных свойств;
(с) одну из последовательностей, образованных вариантами последовательностей (а) и (b).
9) Другой аспект изобретения относится к полипептиду, обладающему по меньшей мере 70%, 80%, 85%, 90%, 95% или 98% идентичности по аминокислотам с полипептидом, включающим по меньшей мере 10, 15, 20, 30-40, 50, 60, 70, 80, 90, 100, 120, 140, 160, 180, 200, 220, 240, 260, 280, 300, 320, 340, 360, 380, 400, 420, 440, 460, 480, 500, 520, 540, 560, 580, 600, 620 или 640 последовательных аминокислот полипептида согласно вышеприведенному п.8).
10) Еще один аспект изобретения относится к полипептиду по вышеприведенному п.9), отличающемуся тем, что он выделен из бактерии рода Streptomyces.
11) Другой аспект изобретения относится к полипептиду по вышеприведенному п.9) или 10), отличающемуся тем, что он кодирует протеин, участвующий в биосинтезе макролида.
12) Следующий аспект изобретения относится к полипептиду по вышеприведенному п.9), 10) или 11), отличающемуся тем, что он обладает активностью, подобной таковой полипептида, с которым он имеет идентичность.
13) Еще один аспект изобретения относится к рекомбинантной ДНК, отличающейся тем, что она включает по меньшей мере один полинуклеотид по любому из вышеприведенных п.п.1), 2), 3), 4), 5) или 6).
14) Другой аспект изобретения относится к рекомбинантной ДНК по вышеприведенному п.13, отличающейся тем, что указанная рекомбинантная ДНК встроена в вектор.
15) Следующий аспект изобретения относится к рекомбинантной ДНК по вышеприведенному п.14, отличающейся тем, что вышеуказанный вектор выбирают среди бактериофагов, плазмид, фагемид, интегрирующих векторов, фосмид, космид, шаттл-векторов, ВАС или РАС.
16) Другой аспект изобретения относится к рекомбинантной ДНК по вышеприведенному п.15, отличающейся тем, что ее выбирают среди pOS49.1, pOS49.11, pOSC49.12, pOS49.14, pOS49.16, pOS49.28, pOS44.1, pOS44.2, pOS44.4, pSPM5, pSPM7, pOS49.67, pOS49.88, pOS49.106, pOS49.120, pOS49.107, pOS49.32, pOS49.43, pOS49.44, pOS49.50, pOS49.99, pSPM17, pSPM21, pSPM502, pSPM504, pSPM507, pSPM508, pSPM509, pSPM1, pBXL1111, pBXL1112, pBXL1113, pSPM520, pSPM521, pSPM522, pSPM523, pSPM524, pSPM525, pSPM527, pSPM528, pSPM34, pSPM35, pSPM36, pSPM37, pSPM38, pSPM39, pSPM40, pSPM41, pSPM42, pSPM43, pSPM44, pSPM45, pSPM47, pSPM48, pSPM50, pSPM51, pSPM52, pSPM53, pSPM55, pSPM56, pSPM58, pSPM72, pSPM73, pSPM515, pSPM519, pSPM74, pSPM75, pSPM79, pSPM83, pSPM107, pSPM543 и pSPM106.
17) Следующий аспект изобретения относится к экспрессирующему вектору, отличающемуся тем, что он включает по меньшей мере одну нуклеотидную последовательность, кодирующую полипептид по вышепривденному п.7), 8), 9), 10), 11) или 12).
18) Изобретение также относится к экспрессионной системе, включающей соответствующий экспрессирующий вектор и клетку-хозяина, позволяющей осуществлять экспрессию одного или нескольких полипептидов по вышеприведенному п.7), 8), 9), 10), 11) или 12.
19) Изобретение также относится к экспрессионной системе по вышеприведенному п.18, отличающейся тем, что ее выбирают из прокариотных экспрессионных систем или эукариотных экспрессирующих систем.
20) Изобретение также относится к экспрессионной системе по вышеприведенному п.19), отличающейся тем, что ее выбирают из экспрессионных систем бактерии E.coli, бакуловирусных экспрессионных систем, позволяющих осуществлять экспрессию в клетках насекомых, экспрессиионных систем, позволяющих осуществлять экспрессию в дрожжевых клетках, экспрессионных систем, позволяющих осуществлять экспрессию в клетках млекопитающих.
21) Изобретение также относится к клетке-хозяину, в которую введен по меньшей мере один полинуклеотид и/или по меньшей мере одна рекомбинантная ДНК и/или по меньшей мере один экспрессирующий вектор по любому из вышеприведенных п.п.1), 2), 3), 4), 5), 6), 13), 14), 15), 16) или 17.
22) Изобретение также относится к способу продуцирования полипептида по вышеприведенному п.7), 8), 9), 10), 11) или 12), отличающемуся тем, что вышеуказанный способ включает следующие стадии:
а) инсерция по меньшей мере одной нуклеиновой кислоты, кодирующей вышеуказанный полипептид, в соответствующий вектор;
b) культивирование, на соответствующей культуральной среде, клетки-хозяина, предварительно трансформированной или трансфектированной вектором со стадии а);
с) рекуперация кондиционированной культуральной среды или клеточного экстракта;
d) выделение и очистка из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ного) на стадии с), вышеуказанного полипептида;
е) в желательном случае, охарактеризовывание продуцированного рекомбинантного полипептида.
23) Другой аспект изобретения относится к микроорганизму, блокируемому на стадии пути биосинтеза по меньшей мере одного макролида.
24) Следующий аспект изобретения относится к микроорганизму по вышеприведенному п.23), отличающемуся тем, что его получают путем инактивации функции по меньшей мере одного протеина, участвующего в биосинтезе этого или этих макролидов в микроорганизме, являющемся продуцентом этого или этих макролидов.
25) Другой аспект изобретения относится к микроорганизму по вышеприведенному п.24), отличающемуся тем, что инактивацию этого протеина или этих протеинов осуществляют путем мутагенеза в гене или генах, кодирующих вышеуказанный или вышеуказанные протеины, или путем экспрессии одной или нескольких антисмысловых РНК, комплементарных РНК или матричным РНК, кодирующим вышеуказанный или вышеуказанные протеины.
26) Другой аспект изобретения относится к микроорганизму по вышеприведенному п.25, отличающемуся тем, что инактивацию этого или этих протеинов осуществляют за счет мутагенеза путем облучения, путем воздействия мутагенного химического агента, путем направленного мутагенеза или путем замены гена.
27) Следующий аспект изобретения относится к микроорганизму по вышеприведенному п.25) или 26), отличающемуся тем, что мутагенез или мутаганезы осуществляют in vitro или in situ путем супрессии, замены, делеции и/или инсерции одного или нескольких оснований в случае рассматриваемого гена или рассматриваемых генов или путем генной инактивации.
28) Еще один аспект изобретения относится к микроорганизму по вышеприведенному п.23), 24), 25), 26) или 27), отличающемуся тем, что вышеуказанным микроорганизмом является бактерия рода Streptomyces.
29) Другой аспект изобретения относится к микроорганизму по вышеприведенному п.23), 24), 25), 26), 27) или 28), отличающемуся тем, что макролидом является спирамицин.
30) Другой аспект изобретения относится к микроорганизму по вышеприведенному п.23), 24), 25), 26), 27), 28) или 29, отличающемуся тем, что вышеуказанным микроорганизмом является штамм S. ambofaciens.
31) Другой аспект изобретения относится к микроорганизму по вышеприведенному п.23), 24), 25), 26), 27), 28), 29) или 30), отличающемуся тем, что мутагенез осуществляют по меньшей мере в гене, включающем последовательность по любому из вышеуказанных п.п.1), 2), 3), 4), 5) или 6).
32) Следующий аспект изобретения относится к микроорганизму по вышеприведенному п.25), 26), 27), 28), 29), 30) или 31, отличающемуся тем, что мутагенез осуществляют в одном или нескольких генах, включающих одну из последовательностей, соответствующих одной или нескольким последовательностям SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 107, 109, 111, 113, 115, 118, 120, 141, 143, 145, 147 и 149.
33) Другой аспект изобретения относится к микроорганизму по вышеприведенному п.25), 26), 27), 28), 29), 30), 31) или 32, отличающемуся тем, что мутагенез состоит в генной инактивации гена, включающего последовательность, соответствующую проследовательности SEQ ID № 13.
34) Другой аспект изобретения относится к штамму Streptomyces ambofaciens, отличающемуся тем, что он является штаммом, выбираемым из одного из штаммов, депонированных в Национальной коллекции культур микроорганизмов (CNCM) 10 июля 2002 г. под регистрационным номером I-2909, I-2911, I-2912, I-2913, I-2914, I-2915, I-2916 или I-2917.
35) Следующий аспект изобретения относится к способу получения медиатора биосинтеза макролида, отличающемуся тем, что он включает следующие стадии:
а) культивирование, на соответствующей культуральной среде, микроорганизма по любому из вышеприведенных п.п.23), 24), 25), 26), 27), 28), 29), 30), 31), 32), 33) или 34);
b) рекуперация кондиционированной культуральной среды или клеточного экстракта;
с) выделение и очистка из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ного) на стадии b), вышеуказанного медиатора биосинтеза.
36) Другой аспект изобретения относится к способу получения молекулы, происходящей от макролида, отличающемуся тем, что получают медиатор биосинтеза согласно способу по вышеприведенному п.35) и модифицируют таким образом продуцированный медиатор.
37) Следующий аспект изобретения относится к способу получения по вышеприведенному п.36), отличающемуся тем, что вышеуказанный медиатор модифицируют химическим, биохимическим, ферментативным и/или микробиологическим способом.
38) Другой аспект изобретения относится к способу получения по вышеприведенному п.36) или 37), отличающемуся тем, что в вышеуказанный микроорганизм вводят один или несколько генов, кодирующих протеины, способные модифицировать медиатор, служащий для них в качестве субстрата.
39) Следующий аспект изобретения относится к способу получения по вышеприведенному п.36), 37) или 38), отличающемуся тем, что макролидом является спирамицин.
40) Другой аспект изобретения относится к способу получения по вышеприведенному п.36), 37), 38 или 39), отличающемуся тем, что используемым микроорганизмом является штамм S. ambofaciences.
41) Следующий аспект изобретения относится к микроорганизму, продуцирующему спирамицин I, но не продуцирующему спиромицины II и III.
42) Другой аспект изобретения относится к микроорганизму по вышеприведенному п.41), отличающемуся тем, что он включает совокупность генов, необходимых в биосинтезе спирамицина I, однако, ген, включающий последовательность SEQ ID № 13 или один из ее вариантов или одну из последовательностей, происходящих от них в соответствии с вырожденностью генетического кода, и кодирующий полипептид, соответствующий последовательности SEQ ID № 14 или одному из ее вариантов, не экспрессирован или инактивирован.
43) Следующий аспект изобретения относится к микроорганизму по вышеприведенному п.42), отличающемуся тем, что вышеуказанную инактивацию осуществляют путем мутагенеза в гене, кодирующем вышеуказанный протеин, или путем экспрессии антисмысловой РНК, комплементарной матричной РНК, кодирующей вышеуказанный протеин.
44) Другой аспект изобретения относится к микроорганизму по вышеприведенному п.42), отличающемуся тем, что вышеуказанный мутагенез осуществляют в промоторе этого гена, в кодирующей последовательности или в некодирующей последовательности с целью инактивации кодируемого протеина или предотвращения его экспрессии или его трансляции.
45) Следующий аспект изобретения относится к микроорганизму по вышеприведенному п.43) или 44), отличающемуся тем, что мутагенез осуществляют путем облучения, путем воздействия мутагенного химического агента, путем направленного мутагенеза или путем замены гена.
46) Другой аспект изобретения относится к микроорганизму по вышеприведенному п.43), 44) или 45), отличающемуся тем, что мутагенез осуществляют in vitro или in situ путем супрессии, замены, делеции и/или инсерции одного или нескольких оснований в случае рассматриваемого гена или путем генной инактивации.
47) Следующий аспект изобретения относится к микроорганизму по вышеприведенному п.41) или 42), отличающемуся тем, что вышеуказанный микроорганизм получают за счет экспрессии генов пути биосинтеза спирамицина, причем они не включают ген, содержащий последовательность, соответствующую SEQ ID № 13, или один из ее вариантов или одну из последовательностей, происходящих от них в соответствии с вырожденностью генетического кода, и кодирующий полипептид, соответствующий последовательности SEQ ID № 14 или одному из ее вариантов.
48) Другой аспект изобретения относится к микроорганизму по вышеприведенному п.41), 42), 43), 44), 45), 46) или 47), отличающемуся тем, что вышеуказанным микроорганизмом является бактерия рода Streptomyces.
49) Следующий аспект изобретения относится к микроорганизму по вышеприведенному п.41), 42), 43), 44), 45) 46), 47) и 48), отличающемуся тем, что вышеуказанный микроорганизм получают из исходного штамма, продуцирующего спирамицины I, II и III.
50) Другой аспект изобретения относится к микроорганизму по вышеприведенному п.41), 42), 43), 44), 45), 46), 47), 48) или 49), отличающемуся тем, что его получают путем мутагенеза в гене, включающем последовательность, соответствующую SEQ ID № 13, или один из ее вариантов или одну из последовательностей, происходящих от них в соответствии с вырожденностью генетического кода, и кодирующем полипептид, соответствующий последовательности SEQ ID № 14 или одному из ее вариантов, обладающему такой же функцией.
51) Следующий аспект изобретения относится к микроорганизму по вышеприведенному п.41), 42), 43), 44), 45), 46), 47), 48), 49) или 50), отличающемуся тем, что вышеуказанный микроорганизм получают из штамма S. ambofaciens, продуцирующего спирамицины I, II и III, в котором осуществляют генную инактивацию гена, включающего последовательность, соответствующую последовательности SEQ ID № 13, или одну из последовательностей, происходящих от нее в соответствии с вырожденностью генетического кода.
52) Другой аспект изобретения относится к штамму S. ambofaciens, отличающемуся тем, что речь идет о штамме, депонированном в Национальной коллекции культур микроорганизмов (CNCM) 10 июля 2002 г. под регистрационным номером I-2910.
53) Следующий аспект изобретения относится к способу продуцирования спирамицина I, отличающемуся тем, что он включает следующие стадии:
а) культивирование, на соответствующей культуральной среде, микроорганизма по любому из вышеприведенных п.п.41), 42), 43), 44), 45), 46), 47), 48), 49), 50), 51) или 52);
b) рекуперация кондиционированной культуральной среды или клеточного экстракта;
с) выделение и очистка из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ного) на стадии b), спирамицина I.
54) Другой аспект изобретения относится к применению полинуклеотида по любому из вышеприведенных п.п.1), 2), 3), 4), 5) или 6) для повышения продуцирования макролидов микроорганизмом.
55) Следующиий аспект изобретения относится к мутантному микроорганизму, являющемуся продуцентом макролида, отличающемуся тем, что он обладает генетической модификацией по меньшей мере в одном гене, включающем последовательность по любому из вышеприведенных п.п.1), 2), 3), 4), 5) или 6), и/или что он сверхпродуцирует по меньшей мере один ген, включающий последовательность по любому из вышеприведенных п.п.1), 2), 3), 4), 5) или 6).
56) Другой аспект изобретения относится к мутантному микроорганизму по вышеприведенному п.55), отличающемуся тем, что генетическая модификация состоит в супрессии, замене, делеции и/или инсерции одного или нескольких оснований в случае рассматриваемого гена или рассматриваемых генов с целью экспрессирования одного или нескольких протеинов, обладающих высшей активностью, или экспрессирования на более высоком уровне этого или этих протеинов.
57) Следующий аспект изобретения относится к мутантному микроорганизму по вышеприведенному п.55), отличающемуся тем, что сверхпродуцирования рассматриваемого гена достигают за счет увеличения числа копий этого гена и/или за счет введения более активного промотора, чем исходный промотор.
58) Другой аспект изобретения относится к мутантному микроорганизму по вышеприведенному п.55) или 57), отличающемуся тем, что сверхпродуцирования рассматриваемого гена достигают за счет трансформации микроорганизма, являющегося продуцентом макролида, с помощью конструкции рекомбинантной ДНК по вышеприведенному п.13, 14 или 17, позволяющей осуществлять сверхпродуцирование этого гена.
59) Следующий аспект изобретения относится к мутантному микроорганизму по вышеприведенному п.55), 56), 57) или 58), отличающемуся тем, что генетическую модификацию осуществляют в одном или нескольких генах, включающих одну из последовательностей, соответствующих одной или нескольким последовательностям SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 107, 109, 111, 113, 115, 118, 120, 141, 143, 145, 147 и 149, или один из ее вариантов, или одну из последовательностей, происходящих от них в соответствии с вырожденностью генетического кода.
60) Другой аспект изобретения относится к мутантному микроорганизму по вышеприведенному п.55), 56), 57), 58) или 59), отличающемуся тем, что микроорганизм сверхпродуцирует один или несколько генов, включающих одну из последовательностей, соответствующих одной или нескольким последовательностям SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 107, 109, 111, 113, 115, 118, 120, 141, 143, 145, 147 и 149, или один из ее вариантов, или одну из последовательностей, происходящих от них в соответствии с вырожденностью генетического кода.
61) Другой аспект изобретения относится к мутантному микроорганизму по вышеприведенному п.55), 56), 57), 58), 59) или 60), отличающемуся тем, что вышеуказанным микроорганизмом является одна из бактерий рода Streptomyces.
62) Следующий аспект изобретения относится к мутантному микроорганизму по вышеприведенному п.55), 56), 57), 58), 59), 60) или 61), отличающемуся тем, что макролидом является спирамицин.
63) Другой аспект изобретения относится к мутантному микроорганизму по вышеприведенному п.55), 56), 57), 58), 59), 60), 61) или 62), отличающемуся тем, что вышеуказанным микроорганизмом является один из штаммов S. ambofaciens.
64) Следующий аспект изобретения относится к способу продуцирования макролидов, отличающемуся тем, что он включает следующие стадии:
а) культивирование, на соответствующей культуральной среде, микроорганизма по любому из вышеприведенных п.п.55), 56), 57), 58), 59), 60), 61), 62), 63) или 64);
b) рекуперация кондиционированной культуральной среды или клеточного экстракта;
с) выделение и очистка из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ного) на стадии b), вышеуказанного продуцированного макролида или вышеуказанных продуцированных макролидов.
65) Другой аспект изобретения относится к применению последовательности и/или рекомбинантной ДНК и/или вектора по любому из вышеприведенных п.п.1), 2), 3), 4), 5), 6), 7), 8), 9), 10), 11), 12), 13), 14), 15), 16) или 17) для получения гибридных антибиотиков.
66) Следующий аспект изобретения относится к применению по меньшей мере одного полинуклеотида и/или по меньшей мере одной рекомбинантной ДНК и/или по меньшей мере одного экспрессирующего вектора и/или по меньшей мере одного полипептида и/или по меньшей мере одной клетки-хозяина по одному из вышеприведенных п.п.1), 2), 3), 4), 5), 6), 7), 8), 9), 10), 11), 12), 13), 14), 15), 16), 17) или 21) для осуществления одной или нескольких биоконверсий.
67) Другой аспект изобретения относится к полинуклеотиду, отличающемуся тем, что речь идет о полинуклеотиде, комплементарном одному из полинуклеотидов по вышеприведенному п.1), 2), 3), 4), 5) или 6).
68) Следующий аспект изобретения относится к микроорганизму, являющемуся продуцентом по меньшей мере одного спирамицина, отличающемуся тем, что он сверхпродуцирует:
- ген, который может быть получен за счет амплификации путем полимеразной цепной реакции (PCR) при использовании пары праймеров, имеющих следующие последовательности: 5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID № 138) и 5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID № 139) и в качестве матрицы космиды pSPM36 или полной ДНК Streptomyces ambofaciens;
- или ген, происходящий от него в соответствии с вырожденностью генетического кода.
69) Другой аспект изобретения относится к микроорганизму по п.68 или 90, отличающемуся тем, что речь идет о бактерии рода Streptomyces.
70) Следующий аспект изобретения относится к микроорганизму по п.68, 69 или 90, отличающемуся тем, что речь идет о бактерии вида Streptomyces ambofaciens.
71) Другой аспект изобретения относится к микроорганизму по п.68, 69, 70 или 90, отличающемуся тем, что сверхпродуцирования вышеуказанного гена достигают путем трансформации вышеуказанного микроорганизма экспрессирующим вектором.
72) Следующий аспект изобретения относится к штамму Streptomyces ambofaciens, отличающемуся тем, что речь идет о штамме OSC2/pSPM75(1) или штамме OSC2/pSPM75(2), депонированном в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 6 октября 2003 г. под регистрационным номером I-3101.
73) Другой аспект изобретения относится к рекомбинантной ДНК, отличающейся тем, что она включает:
- полинуклеотид, который может быть получен за счет амплификации путем полимеразной цепной реакции при использовании пары праймеров, имеющих следующие последовательности: 5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID № 138) и 5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID № 139) и в качестве матрицы космиды pSPM36 или полной ДНК Streptomyces ambofaciens;
- или фрагмент, включающий по меньшей мере 10, 12, 15, 18, 20-25, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 350, 400, 450, 500, 550, 600, 650, 700, 750, 800, 850, 900, 950, 1000, 1050, 1100, 1150, 1200, 1250, 1300, 1350, 1400, 1450, 1460, 1470, 1480, 1490 или 1500 последовательных нуклеотидов этого полинуклеотида.
74) Следующий аспект изобретения относится к рекомбинантной ДНК по п.73 или 91, отличающейся тем, что речь идет о векторе.
75) Другой аспект изобретения относится к рекомбинантной ДНК по п.73, 74 или 91, отличающейся тем, что речь идет о экспрессирующем векторе.
76) Следующий аспект изобретения относится к клетке-хозяину, в которую введена по меньшей мере одна рекомбинантная ДНК по любому из п.п.73, 74, 75 или 91.
77) Другой аспект изобретения относится к способу продуцирования полипептида, отличающемуся тем, что вышеуказанный способ включает следующие стадии:
а) трансформация клетки-хозяина с помощью по меньшей мере одного экспрессирующего вектора по п.75;
b) культивирование, на соответствующей культуральной среде, вышеуказанной клетки-хозяина;
с) рекуперация кондиционированной культуральной среды или клеточного экстракта;
d) выделение и очистка из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ного) на стадии с), вышеуказанного полипептида;
е) в желательном случае, охарактеризовывание продуцированного рекомбинантного полипептида.
78) Следующий аспект изобретения относится к микроорганизму по п.51, отличающемуся тем, что генную инактивацию осуществляют путем делеции полноразмерного гена или части гена, включающего последовательность, соответствующую SEQ ID № 13, или одну из последовательностей, происходящих от нее в соответствии с вырожденностью генетического кода.
79) Другой аспект изобретения относится к микроорганизму по любому из п.п.41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51 или 78, отличающемуся тем, что он, кроме того, сверхпродуцирует:
- ген, который может быть получен за счет амплификации путем полимеразной цепной реакции при использовании пары праймеров, имеющих следующие последовательности: 5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID № 138) и 5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID № 139) и в качестве матрицы космиды pSPM36 или полной ДНК Streptomyces ambofsciens;
- или ген, происходящий от него в соответствии с вырожденностью генетического кода.
80) Следующий аспект изобретения относится к экспрессирующему вектору, отличающемуся тем, что полинуклеотид, соответствующий последовательности SEQ ID № 47, или полинуклеотид, происходящий от него в соответствии с вырожденностью генетического кода, находится под контролем промотора, позволяющего осуществлять экспрессию протеина, кодируемого указанным полинуклеотидом, в Streptomyces ambofaciens.
81) Другой аспект изобретения относится к экспрессирующему вектору по п.80, отличающемуся тем, что он является плазмидой pSPM524 или pSPM525.
82) Следующий аспект изобретения относится к штамму Streptomyces ambofaciens, трансформированному вектором по п.80 или 81.
83. Другой аспект изобретения относится к микроорганизму по любому из п.п.41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 78, 79 или 92, отличающемуся тем, что он, кроме того, сверхпродуцирует ген, соответствующий кодирующей последовательности SEQ ID 47 или кодирующей последовательности, происходящей от нее в соответствии с вырожденностью генетического кода.
84. Следующий аспект изобретения относится к микроорганизму по п.83, отличающемуся тем, что речь идет о штамме SPM502pSPM525, депонированном в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 26 февраля 2003 г. под регистрационным номером I-2977.
85) Другой аспект изобретения относится к способу продуцирования спирамицина (спирамицинов), отличающемуся тем, что он включает следующие стадии:
а) культивирование, на соответствующей культуральной среде, микроорганизма по любому из п.п.68, 69, 70, 71, 72, 78, 79, 82, 83, 84, 90 или 92;
b) рекуперация кондиционированной культуральной среды или клеточного экстракта;
с) выделение и очистка из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ного) на стадии b), спирамицинов.
86) Следующий аспект изобретения относится к полипептиду, отличающемуся тем, что его последовательность включает последовательность SEQ ID № 112 или последовательность SEQ ID № 142.
87) Другой аспект изобретения относится к полипептиду, отличающемуся тем, что его последовательность соответствует последовательности, выражаемой кодирующей последовательностью:
- гена, который может быть получен за счет амплификации путем полимеразной цепной реакции (РСR) при использовании пары праймеров, имеющих следующие последовательности: 5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID № 138) и 5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID № 139) и в качестве матрицы космиды pSPM36 или полной ДНК Streptomyces ambofaciens,
- или гена, происходящего от него в соответствии с вырожденностью генетического кода.
88) Следующий аспект изобретения относится к экспрессирующему вектору, позволяющему осуществлять экспрессию полипептида по п.86, 87 или 93 в Streptomyces ambofaciens.
89) Другой аспект изобретения относится к экспрессирующему вектору по п.88, отличающемуся тем, что речь идет о плазмиде pSPM75.
90) Следующий аспект изобретения относится к микроорганизму по п.68, отличающемуся тем, что геном, который может быть получен за счет амплификации путем полимеразной цепной реакции, является ген, соответствующий кодирующей последовательности SEQ ID № 141, или ген, происходящий от него в соответствии с вырожденностью генетического кода.
91) Другой аспект изобретения относится к рекомбинантной ДНК по п.73, отличающейся тем, что полинуклеотидом, который может быть получен за счет амплификации путем полимеразной цепной реакции, является полинуклеотид, соответствующий последовательности SEQ ID № 141.
92) Следующий аспект изобретения относится к микроорганизму по п.79, отличающемуся тем, что геном, который может быть получен за счет амплификации путем полимеразной цепной реакции, является ген, соответствующий кодирующей последовательности SEQ ID № 141, или ген, происходящий от него в соответствии с вырожденностью генетического кода.
93) Другой аспект изобретения относится к полипептиду, отличающемуся тем, что его последовательностью является SEQ ID № 142.
Общие определения
Термин “выделенный” в смысле настоящего изобретения означает биологический материал (нуклеиновая кислота или протеин), который извлекают из его первоначальной окружающей среды (окружающая среда, в которой он локализован естественным образом).
Например, полинуклеотид, присутствующий в нативном состоянии в растении или животном, не является выделенным. Тот же полинуклеотид, отделенный прилегающими нуклеиновыми кислотами, внутри которых он естественным образом встроен в геном растения или животного, рассматривают как “выделенный”.
Такой полинуклеотид может быть встроен в вектор и/или такой полинуклеотид может быть включен в состав и тем не менее находится в выделенном состоянии из-за того, что вектор или состав не образует его нативную окружающую среду.
Термин “очищенный” не требует того, чтобы материал находился в абсолютно чистой форме, исключающей присутствие других соединений. Скорее речь идет об относительном определении.
Полинуклеотид находится в “чистом” состоянии после очистки исходного материала или нативного материала по меньшей мере одного порядка величины, предпочтительно 2 или 3 и особенно предпочтительно 4 или 5 порядков величины.
Согласно настоящему изобретению термин ORF (“Open Reading Frame”, то есть открытая рамка считывания) используют для обозначения, в частности, кодирующей последовательности гена.
Согласно настоящему описанию выражение “нуклеотидная последовательность” может быть использовано для обозначения безразлично полинуклеотида или нуклеиновой кислоты. Выражение “нуклеотидная последовательность” включает сам генетический материал и, следовательно, не является ограниченным информацией, касающейся его последовательности.
Термины “нуклеиновая кислота”, “полинуклеотид”, “олигонуклеотид” или еще “нуклеотидная последовательность” включают последовательности РНК, ДНК, кДНК или еще гибридные последовательности РНК/ДНК из более чем одного нуклеотида, безразлично в виде ординарной цепи или в виде двойной цепи.
Термин “нуклеотид” означает одновременно природные нуклеотиды (A, T, G, C), а также модифицированные нуклеотиды, которые включают по меньшей мере одну модификацию, такую, как (1) аналог пурина, (2) аналог пиримидина или (3) аналог сахара, причем промеры таких модифицированных нуклеотидов описываются, например в заявке РСТ № WO-95/04064.
Согласно настоящему изобретению первый полинуклеотид рассматривают как “комплементарный” второму полинуклеотиду, тогда как каждое основание первого полинуклеотида “спарено” с комплементарным основанием второго полинуклеотида, ориентация которого является обратной. Комплементарными основаниями являются А и Т (или А и U) или С и G.
Термин “гены пути биосинтеза спирамицинов” также включает гены-регуляторы и гены, придающие резистентность к микроорганизмам-продуцентам.
Под “фрагментом” стандартной нуклеиновой кислоты согласно изобретению понимают нуклеотидную последовательность уменьшенной длины по отношению к стандартной нуклеиновой кислоте и включающую, в общей части, нуклеотидную последовательность, идентичную стандартной нуклеиновой кислоте.
Такой “фрагмент” нуклеиновой кислоты согласно изобретению, в желательном случае, может быть встроен в полинуклеотид большей величины, для которого он является конститутивным.
Такие фрагменты включают или, альтернативно, состоят из полинуклеотидов длины, доходящей до 8, 10, 12, 15, 18, 20-25, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 350, 400, 450, 500, 550, 600, 650, 700, 750, 800, 850, 900, 950, 1000, 1050, 1100, 1150, 1200, 1250, 1300, 1350, 1400, 1450, 1500, 1550, 1600, 1650, 1700, 1750, 1800, 1850 или 1900 последовательных нуклеотидов нуклеиновой кислоты согласно изобретению.
Под “фрагментом” полипептида согласно изобретению понимают полипептид, аминокислотная последовательность которого более короткая, чем таковая стандартного полипептида, и который включает, в целом, часть, общую с этими стандартными полипептидами, идентичной аминокислотной последовательности.
Такие фрагменты, в желательном случае, могут быть встроены в полипептид большей величины, частью которого они являются.
Фрагменты полипептида согласно изобретению могут иметь длину, составляющую 10, 15, 20, 30-40, 50, 60, 70, 80, 90,100, 120, 140, 160, 180, 200, 220, 240, 260, 280, 300, 320, 340, 360, 380, 400, 420, 440, 460, 480, 500, 520, 540, 560, 580, 600, 620 или 640 аминокислот.
Под “условиями гибридизации высокой жесткости” согласно настоящему изобретению понимают условия гибридизации, неблагоприятные для гибридизации нитей негомологичных нуклеиновых кислот. Условия гибридизации высокой жесткости могут быть описаны, например, как условия гибридизации в буфере, описанном Church и Gilbert (Church & Gilbert, 1984), при температуре от 55°С до 65°С, причем, предпочтительно, температура гибридизации составляет 55°С, еще более предпочтительно, температура гибридизации составляет 60°С и, в высшей степени предпочтительно, температура гибридизации составляет 65°С, с последующими одной или несколькими промывками, осуществляемыми в буфере 2Х SSC (буфер 1Х SSC соответствует водному растору 0,15 М NaCl, 15 мМ цитрата натрия), при температуре от 55°С до 65°С, причем, предпочтительно, эта температура составляет 55°С, еще более предпочтительно, эта температура составляет 60°С и, в высшей степени предпочтительно, эта температура составляет 65°С, с последующими одной или несколькими промывками, осуществляемыми в буфере 0,5Х SSC при температуре от 55°С до 65°С, причем, предпочтительно, эта температура составляет 55°С, еще более предпочтительно, эта температура составляет 60°С и, в высшей степени предпочтительно, эта температура составляет 65°С.
Само собой разумеется, что вышеописанные условия гибридизации могут быть адаптированы в зависимости от длины нуклеиновой кислоты, гибридизацию которой стремятся осуществить, или выбранного типа маркировки, согласно способам, известным специалисту в данной области. Соответствующие условия гибридизации могут быть адаптированы, например, согласно работе F. Ausubel и др. (2002).
Под “вариантом” нуклеиновой кислоты согласно изобретению понимают нуклеиновую кислоту, которая отличается одним или несколькими основаниями по сравнению со стандартным полинуклеотидом. Вариант нуклеиновой кислоты может быть природного происхождения, такой, как естественно встречающийся аллельный вариант, или также может представлять собой не нативный вариант, получаемый, например, способами мутагенеза.
Вообще, различия между стандартной нуклеиновой кислотой и вариантом нуклеиновой кислоты ограничены таким образом, что нуклеотидные последовательности стандартной нуклеиновой кислоты и варианта нуклеиновой кислоты являются очень близкими и в многочисленных областях идентичными. Модификации нуклеотидов, находящихся в варианте нуклеиновой кислоты, могут быть молчащими, что означает, что они не изменяют аминокислотные последовательности, кодируемые вышеуказанным вариантом нуклеиновой кислоты.
Однако изменения нуклеотидов в варианте нуклеиновой кислоты также могут происходить в результате замен, инсерций, делеций в полипептиде, кодируемом вариантом нуклеиновой кислоты, по сравнению с пептидами, кодируемыми стандартной нуклеиновой кислотой. Кроме того, модификации нуклеотидов в кодирующих областях могут продуцировать консервативные или неконсервативные замены в аминокислотной последовательности.
Предпочтительно, варианты нуклеиновых кислот согласно изобретению кодируют полипептиды, которые в значительной степени сохраняют ту же самую функцию или биологическую активность, как полипептид, соответствующий стандартной нуклеиновой кислоте, или еще способность распознаваться антителами, направленными против полипептидов, кодируемых первоначальной нуклеиновой кислотой.
Некоторые варианты нуклеиновых кислот кодируют также и мутированные формы полипептидов, систематическое исследование которых позволит сделать вывод о связях структуры с активностью соответствующих протеинов.
Под “вариантом” полипептида согласно изобретению понимают, в принципе, полипептид, аминокислотная последовательность которого содержит одну или несколько замен, инсерций или делеций по меньшей мере одного аминокислотного остатка по сравнению с аминокислотной последовательностью стандартного полипептида, имея в виду, что замены аминокислот могут быть, безразлично, консервативными или неконсервативными.
Предпочтительно, полипетидные варианты согласно изобретению в значительной степени сохраняют ту же самую функцию или биологическую активность, как стандартный полипептид, или еще способность распознаваться антителами, направленными против первоначальных полипептидов.
Под полипептидом, “обладающим активностью, подобной” стандартному полипептиду, согласно изобретению понимают полипептид, обладающий близкой биологической активностью, но необязательно идентичной таковой стандартного полипептида, такой, как оцениваемый в соответствующем биологическом тесте для определения биологической активности стандартного полипептида.
Под “гибридным антибиотиком” согласно изобретению понимают соединение, генерируемое искусственной конструкцией пути биосинтеза при использовании технологии рекомбинантной ДНК.
Подробное описание изобретения
Объектом настоящего изобретения в особенности являются новые гены пути биосинтеза спирамицинов и новые полипептиды, участвующие в этом биосинтезе, такие, как представленные в нижеприводимом подробном описании.
Гены пути биосинтеза клонировали и определяли последовательность ДНК этих генов. Полученные последовательности анализировали по программе FramePlot (Ishikawa J. & Hotta K., 1999). Среди открытых рамок считывания идентифицировали открытые рамки считывания, обладающие типичными кодонами Streptomyces. Этот анализ показал, что эта область включает 44 открытые рамки считывания (ORFs), фланкированные с той и другой стороны пятью генами, кодирующими фермент “поликетидсинтазу” (PKS) и обладающие типичными кодонами Streptomyces. С той и другой стороны этих пяти генов, кодирующих PKS, идентифицировали, соответственно, 10 и 34 ORFs ниже и выше (низ и верх определяются ориентацией 5 генов, которые все имеют одну и ту же смысловую ориентацию) (см. фиг.3 и 37). Таким образом, 34 открытые рамки считывания этого типа, занимающие область около 41,7 т.н. (см. SEQ ID № 1, представляющую собой первую область длиной 31 т.н., содержащую 25 ORFs, и SEQ ID № 140, представляющую собой область длиной около 12,1 т.н., 1,4 т.н. которой перекрывают предыдущую последовательность (SEQ ID № 1) и около 10,7 т.н. которой соответствуют продолжению последовательности, причем эта последняя часть длиной около 10,7 т.н., содержащая 9 дополнительных открытых рамок считывания (в том числе открытую рамку считывания (ORF) частичной последовательности), см. также фиг.3 и 37 ниже), были идентифицированы выше 5 генов, кодирующих PKS, и 10 открытых рамок считывания, занимающих область около 11,1 т.н. (SEQ ID № 2 и фиг.3), были идентифицированы ниже генов PKS. 10 Генов, расположенных ниже 5 генов PKS, таким образом, были названы orf1*c, orf2*c, orf3*c, orf4*c, orf5*, orf6*, orf7*c, orf8*, orf9*, orf10* (SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19 и 21). Буква “с”, добавленная в название гена означает, для соответствующей рамки считывания, что кодирующая последовательность находится в обратной ориентации (кодирующей нитью, следовательно, является нить, комплементарная последовательности, представленной в виде SEQ ID № 2, этих генов). Используя ту же самую номенклатуру, 34 рамки считывания, находящиеся выше генов PKS, были названы orf1, orf2, orf3, orf4, orf5, orf6, orf7, orf8, orf9с, orf10, orf11с, orf12, orf13с, orf14, orf15с, orf16, orf17, orf18, orf19, orf20, orf21с, orf22с, orf23с, orf24с, orf25с, orf26, orf27, orf28с, orf29, orf30с, orf31, orf32с, orf33 и orf34с (SEQ ID № 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 107, 109, 111, 113, 115, 118, 120, 141, 143, 145, 147 и 149) (см. фиг.3 и 37).
Протеиновые последовательности, следующие из этих открытых рамок считывания, сравнивали с таковыми, имеющимися в различных базах данных благодаря различным программам: BLAST (Altschul и др., 1990; Altschul и др., 1997), CD-search, COGs (Кластер ортологичных групп) (эти три программы доступны, в частности, из NCBI (Национальный центр информации по биотехнологии) (Bethesda, Maryland, США)), FASTA ((Pearson W. R. & Lipman D. J., 1988) и (Pearson W. R., 1990)), BEATY (Worley K. C. и др., 1995) (эти две программы доступны, в частности, из центра ресурсов INFOBIOGEN, Evry, Франция). Эти сравнения позволили сформулировать гипотезы в отношении функции вырабатываемых этими генами продуктов и идентифицировать таковые, способные участвовать в биосинтезе спирамицинов.
Гены, расположенные ниже генов, кодирующих PKS
Схематическое изображение структуры области представлено на фиг.3. Таким образом, как будет показано ниже, из 10 генов, идентифицированных ниже генов, кодирующих PKS, 9 оказываются принимающими участие в биосинтезе или резистентности к спирамицинам. Речь идет о 9 следующих генах: orf1*c, orf2*c, orf3*c, orf4*c, orf5*, orf6*, orf7*c, orf8* и orf9*.
В следующей таблице 1 представлены ссылки на последовательность из ДНК и из аминокислот 10 генов, идентифицированных ниже 5 генов PKS.
Буква “с”, добавленная в название гена, означает, что кодирующая последовательность находится в обратной ориентации (кодирующей нитью, следовательно, является нить, комплементарная последовательности, представленной в виде SEQ ID № 2, этих генов).
С целью определения функции идентифицированных полипептидов были проведены три типа экспериментов: сравнение идентифицированных последовательностей с последовательностями, обладающими известными функциями; эксперименты по инактивации генов, приводящие к конструкции мутантных штаммов; и анализы в отношении продуцирования этими мутантными штаммами спирамицинов и медиаторов биосинтеза спирамицинов.
Протеиновые последовательности, следующие из этих открытых рамок считывания, все сначала сравнивали с таковыми, имеющимися в различных банках данных благодаря различным программам: BLAST (Altschul и др., 1990; Altschul и др., 1997), CD-search, COGs (Кластер ортологичных групп), FASTA ((Pearson W. R. & Lipman D. J., 1988) и (Pearson W. R., 1990)), BEATY (Worley K. C. и др., 1995). Эти сравнения позволили сформулировать гипотезы в отношении функции вырабатываемых этими генами продуктов и идентифицировать таковые, способные принимать участие в биосинтезе спирамицина. В таблице 2 представлены протеины, обладающие большим сходством с продуктами, вырабатываемыми 10 генами, расположенными ниже 5 генов PKS.
Эксперименты по инактивации генов осуществляли для подтверждения этих результатов. Используемые способы состоят в осуществлении замены гена. Прерываемый ген-мишень заменяли копией этого гена, прерванного кластером, придающим резистентность к антибиотику (как, например, апрамицин, генетицин и гигромицин). Используемые кластеры фланкированы с той и другой стороны кодонами терминации трансляции во всех открытых рамках считывания и активными терминаторами транскрипции в Streptomyces. Инсерция кластера в ген-мишень может сопровождаться или нет делецией в этом гене-мишени. Величина областей, фланкирующих кластер, может доходить от нескольких сотен до нескольких тысяч пар оснований. Для инактивации генов может быть использован второй тип кластеров: кластеры, называемые “эксцизируемыми кластерами”. Эти кластеры обладают тем преимуществом, что могут быть эксцизированы в случае Streptomyces за счет явления рекомбинации специфического сайта после того, как были встроены в геном S. ambofaciens. Целью является инактивация некоторых генов в штаммах Streptomyces, не оставляя в конечном штамме маркеров селекции или больших последовательностей ДНК, не принадлежащих штамму. После эксцизии в геноме штамма имеется только короткая последовательность из тридцати пар оснований (называемая “рубцовым” сайтом) (см. фиг.10). Осуществление этой системы состоит, во-первых, в замене копии дикого типа гена-мишени (за счет двух явлений гомологичной рекомбинации, см. фиг.9) конструкцией, в которой эксцизируемый кластер встроен в этот ген-мишень. Инсерции этого кластера сопутствует делеция в гене-мишени (см. фиг.9). Во-вторых, провоцируют эксцизию эксцизируемого кластера из генома штамма. Эксцизируемый кластер функционирует благодаря системе сайтспецифической рекомбинации и обладает преимуществом в том, что позволяет получать мутанты Streptomyces, не содержащие, в конечном счете, гена резистентности. Также преодолеваются возможные полярные воздействия на экспрессию генов, находящихся ниже инактивированного гена или инактивированных генов (см. фиг.10). Таким образом сконструированные штаммы тестировали на продуцирование ими спирамицинов.
Инактивация генов orf1*c, orf2*c, orf3*c и orf4*c не была осуществлена, так как эксперименты в отношении сравнения последовательности позволили определить, что эти гены имеют относительно большое сходство с генами, участвующими в биосинтезе относительно близкого антибиотика. Так, ген orf1* кодирует протеин, имеющий идентичность 66% (определенную согласно программе BLAST) с протеином, кодируемым геном tylMI, который кодирует N-метилтрансферазу, участвующую в биосинтезе тилозина и катализирующую N-метилирование во время продуцирования микаминозы в случае Streptomyces fradiae (Gandecha A. R. и др., 1997; номер доступа в банк генов: САА57473; оценка по BLAST: 287). Это сходство с протеином, участвующим в пути биосинтеза другого, относительно близкого антибиотика и в особенности в биосинтезе микаминозы, позволяет полагать, что ген orf1*c кодирует N-метилтрансферазу, ответственную за N-метилирование во время биосинтеза форозамина или микаминозы (см. фиг.5 и 6). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf1*c, имеет большое сходство с другими протеинами подобной функции в других организмах (см. таблицу 3).
Ген orf2*c кодирует протеин, имеющий относительно большое сходство (35% идентичности) с протеином, кодируемым геном tylMIII, кодирующим NDP-гексозо-3,4-изомеразу, участвующую в биосинтезе тилозина в случае Streptomycts fradiae (Gandecha A. R. и др., 1997; номер доступа в банк генов: САА57471; оценка по BLAST: 130). Это сходство с протеином, участвующим в пути биосинтеза другого близкого антибиотика и, в частности, в биосинтезе микаминозы, позволяет полагать, что ген orf2*c кодирует NDP-гексозо-3,4-изомеразу, ответственную за изомеризацию во время биосинтеза одного из сахаров спирамицина, в известных случаях, микаминозы (см. фиг.5 и 6).
Ген orf3*с кодирует протеин, имеющий относительно большое сходство (59% идентичности) с протеином, кодируемым геном tylMII, который кодирует гликозилтрансферазу, участвующую в биосинтезе тилозина в случае Streptomyces fradiae (Gandecha A. R. и др., 1997; номер доступа в банк генов: САА57472; оценка по BLAST: 448). Это сходство с протеином, участвующим в пути биосинтеза другого близкого антибиотика, позволяет полагать, что ген orf3*c кодирует гликозилтрансферазу. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf3*c, имеет большое сходство с другими протеинами подобной функции в случае других организмов (см. таблицу 4).
Ген orf4*с кодирует протеин, имеющий большое сходство с несколькими кротонил-СоА-редуктазами. В частности, протеин, кодируемый геном orf4*c, обладает значительным сходством с кротонил-СоА-редуктазой в случае Streptomyces coelicolor (Redenbach M. и др., 1996; номер доступа в банк генов: NP_630556; оценка по BLAST: 772). Это сходство с протеином, участвующим в пути биосинтеза другого близкого антибиотика, в высокой степени позволяет полагать, что ген orf4*c кодирует также кротонил-СоА-редуктазу. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf4*c, имеет большое сходство с другими протеинами подобной функции в случае других организмов (см. таблицу 5).
Ген orf6* обладает большим сходством с геном mdmB, имеющимся в случае Streptomyces mycarofaciens (Hara и Hutchisnson, 1992; номер доступа в банк генов: А42719; оценка по BLAST: 489), являющегося продуцентом макролидного антибиотика. В случае этого продуцента ген участвует в ацилировании лактонового цикла. Ген orf6*, следовательно, кодирует ацилтрансферазу. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf6*, имеет большое сходство с другими протеинами подобной функции в случае других организмов (см. таблицу 6).
Инактивацию гена orf6* осуществляли путем делеции/инсерции открытой рамки считывания и показано, что полученный в результате штамм более не продуцирует спирамицин II и спирамицин III, а продуцирует только спирамицин I (см. фиг.1). Это подтверждает то, что ген orf6* действительно участвует в синтезе спирамицина II и спирамицина III. Фермент, кодируемый этим геном, является ответственным за образование спирамицинов II и III путем фиксации ацетильной или бутирильной группы на углероде 3. Штаммы, не экспрессирующие больше протеин, кодируемый геном orf6*, представляют особый интерес, поскольку они не продуцируют больше спирамицины II и III, а продуцируют только спирамицин I. Как указано выше, антибиотическая активность спирамицина I отчетливо выше таковой спирамицинов II и III (Liu и др., 1999).
Ген orf5* кодирует протеин, обладающий относительно большим сходством с несколькими О-метилтрансферазами. В частности, протеин, кодируемый orf5*, обладает значительным сходством с О-метилтрансферазой (ЕС 2.1.1.-) MdmC Streptomyces mycarofaciens (Hara & Hutchinson, 1992; номер доступа в банк генов: В42719; оценка по BLAST: 355). Это сходство с протеином, участвующим в пути биосинтеза другого антибиотика, в высшей степени позволяет полагать, что ген orf5* также кодирует О-метилтрансферазу. Ген orf5* участвует в образовании предшественников, включенных в лактоновый цикл. В самом деле, согласно сравнениям последовательностей, вырабатываемый геном orf5* продукт также относительно близок FkbG, который является ответственным за метилирование гидроксималонил-АСР (согласно Wu и др., 2000; Hoffmeister и др., 2000; номер доступа в банк генов: ААF86386; оценка по BLAST: 247) (см. фиг.8). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf5*, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 7).
Благодаря полярному эффекту инсерции неэксцизированного кластера в гене orf6* определено, что ген orf5* является существенным в пути биосинтеза спирамицинов. В самом деле, инсерция эксцизируемого кластера в кодирующую часть гена orf6* вызывает полное прекращение продуцирования спирамицинов. Однако в случае, когда встроенный кластер эксцизирован (и, следовательно, когда один ген orf6* инактивирован, см. примеры 14 и 15), обнаруживают продуцирование спирамицина I. Это показывает, что ген orf5* является существенным в пути биосинтеза спирамицинов, поскольку его инактивация вызывает полное прекращение продуцирования спирамицинов.
Ген orf5* кодирует протеин, имеющий относительно большое сходство с несколькими О-метилтрансферазами. Ген orf5* является О-метилтрансферазой, участвующей либо непосредственно в синтезе платенолида, либо в синтезе метилированного предшественника (метоксималонил, см. фиг.8), включенного в платенолид с помощью PKS. Для подтверждения этой гипотезы эксперименты с анализом CL/SM (жидкостная хроматография/масс-спектрометрия) и ЯМР осуществляли при использовании штамма S.ambofaciens генотипа orf6*::att1Ωhyg+. В этом штамме ген orf5* не экспрессируется из-за полярного эффекта инсерции, в гене orf6*, кластера, который содержит терминаторы транскрипции (см. пример 27). Показано, что этот штамм продуцирует молекулу, УФ-спектр которой имеет вид, подобный таковому спирамицина I, однако, масс-спектр показывает молекулярный ион 829. Разница массы на 14 по отношению к массе спирамицина может объясняться отсутствием метила у кислорода, находящегося у углерода № 4 лактонового цикла (структура этого соединения представлена на фиг.39). Результаты, полученные с помощью ЯМР, совместимы с этой гипотезой. Наличие соединения с молекулярным ионом 829 позволяет признать действительной гипотезу о роли orf5* в биосинтезе спирамицинов. Кроме того, продукт, соответствующий спирамицину без метильной группы, обладает очень слабой микробиологической активностью (более незначительной на фактор 10) по сравнению с немодифицированным спирамицином, когда тест проводят при использовании микроорганизма Micrococcus luteus.
Ген orf7*с кодирует протеин, имеющий относительно большое сходство с протеином, кодируемым геном mdmA Streptomyces mycarofaciens, причем этот последний кодирует протеин, участвующий в резистентности к мидекамицину в случае этого организма-продуцента (Hara и др., 1990; номер доступа в банк генов: А60725; оценка по BLAST: 380). Это сходство с протеином, участвующим в пути биосинтеза другого антибиотика, в высшей степени позволяет полагать, что ген orf7*с кодирует также протеин, участвующий в резистентности к спирамицину. В особенности, фермент, кодируемый геном orf7*с, обладает метилтрансферазной активностью и участвует в резистентности к спирамицину в случае Streptomyces ambofaciens. Показано, что этот ген придает резистентность типа MLS 1, резистентность, о которой известно, что она возникает вследствие монометилирования в положении А2058 рибосомальной РНК 23S (Pernodet и др., 1996). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf7*с, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 8).
Ген orf8* кодирует протеин, имеющий относительно большое сходство с протеином типа переносчика АВС в Streptomyces griseus (Campelo, 2002; номер доступа в банк генов: САС22119; оценка по BLAST: 191). Это сходство с протеином типа переносчика АВС в высшей степени позволяет полагать, что ген orf8* кодирует также протеин типа переносчика АВС, который может участвовать в резистентности к спирамицину. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf8*, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 9).
Ген orf9* кодирует протеин, имеющий относительно большое сходство с протеином типа переносчика АВС в случае Streptomyces griseus (Campelo, 2002; номер доступа в банк генов: САС22118; оценка по BLAST: 269). Это сходство с протеином типа переносчика АВС в высшей степени позволяет полагать, что ген orf9* кодирует также протеин типа переносчика АВС, который может участвовать в резистентности к спирамицину. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf9*, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 10).
Ген orf10* кодирует протеин, имеющий относительно большое сходство с протеином неизвестной функции. Однако гены, подобные orf10*, обнаружены среди нескольких групп генов, участвующих в биосинтезе антибиотиков. Так, ген, близкий к orf10*, найден в случае S.coelicolor (Redenbach и др., 1996; номер доступа в банк генов: NP_627432; оценка по BLAST: 109). Близкий ген (CouY) также обнаружен в случае S.rishiriensis (Wang и др., 2000; номер доступа в банк генов: ААG29779; оценка по BLAST: 97).
Гены, расположенные выше генов, кодирующих PKS
В последовательности ДНК, расположенной выше генов, кодирующих PKS (низ и верх определяются ориентацией 5 генов, которые все имеют одну и ту же смысловую ориентацию) (см. фиг.3), идентифицированы 34 открытые рамки считывания (см. выше). Таким образом, 34 открытые рамки считывания (ORFs) этого типа, занимающие область около 41,7 т.н. (см. SEQ ID № 1, представляющую собой первую область длиной 31 т.н., содержащую 25 ORFs, и SEQ ID № 140, представляющую собой область длиной около 12,1 т.н., 1,4 т.н. которой перекрывают предыдущую последовательность (SEQ ID № 1) и около 10,7 т.н. которой соответствуют продолжению последовательности), причем эта последняя часть длиной около 10,7 т.н., содержащая 9 дополнительных рамок считывания (в том числе рамкe считывания (ORF) частичной последовательности), см. также фиг.3 и 37 ниже), были идентифицированы выше 5 генов, кодирующих PKS. Схематическое изображение организации области представлено на фиг.3 и 37. 34 Идентифицированные гена были названы: orf1, orf2, orf3, orf4, orf5, orf6, orf7, orf8, orf9с, orf10, orf11с, orf12, orf13с, orf14, orf15с, orf16, orf17, orf18, orf19, orf20, orf21с, orf22с, orf23с, orf24с, orf25с, orf26, orf27, orf28с, orf29, orf30с, orf31, orf32с, orf33 и orf34с.
В следующей таблице 11 представлены обозначения последовательности из ДНК и аминокислот 34 идентифицированных генов, расположенных выше 5 генов PKS.
7534-6686
SEQ ID № 143
SEQ ID № 144
2: Когда для одной рамки считывания (orf) указано несколько протеиновых последовательностей, соответствующие протеины происходят от нескольких возможных сайтов начала трансляции
С целью определения функции полипептидов, идентифицированных согласно вышеприведенной таблице 11, осуществляли три типа экспериментов: сравнение идентичности идентифицированных последовательностей с последовательностями с известными функциями, эксперименты по инактивации генов и анализы продуцирования спирамицинов этими мутантными штаммами.
Протеиновые последовательности, следующие из этих открытых рамок считывания, сначала сравнивали с таковыми, имеющимися в различных базах данных благодаря различным программам: BLAST (Altschul и др., 1990; Altschul и др., 1997), CD-search, COGs (Кластер ортологичных групп), FASTA ((Pearson W. R. & Lipman D. J., 1988) и (Pearson W. R., 1990)), BEATY (Worley K. C. и др., 1995) (см. выше). Эти сравнения позволили сформулировать гипотезы в отношении функции вырабатываемых этими генами продуктов и идентифицировать таковые, способные участвовать в биосинтезе спирамицинов. В таблице 12 представлены протеины, обладающие большим сходством с 34 генами, расположенными выше 5 генов PKS.
Эксперименты по инактивации генов осуществляли для подтверждения этих результатов. Используемые способы состоят в осуществлении замены гена. Прерываемый ген-мишень заменяли копией этого гена, прерванного кластером, придающим резистентность к антибиотику (как, например, апрамицин или гигромицин). Используемые кластеры фланкированы с той и другой стороны кодонами терминации трансляции во всех рамках считывания и активными терминаторами транскрипции в случае Streptomyces. Инсерция кластера в ген-мишень может сопровождаться или нет делецией в этом гене-мишени. Размер областей, фланкирующих кластер, может доходить от нескольких сотен до нескольких тысяч пар оснований. Для инактивации генов может быть использован второй тип кластеров: кластеры, называемые “эксцизируемыми кластерами” (см. выше). Таким образом сконструированные штаммы тестировали на продуцирование ими спирамицинов.
Ген orf1 кодирует протеин, обладающий относительно большим сходством с несколькими цитохромами Р450. В частности, протеин, кодируемый геном orf1, обладает значительным сходством с протеином, кодируемым геном tylI, участвующим в биосинтезе тилозина в Streptomyces fradiae (Merson-Davies L. A. и др., 1994; номер доступа в банк генов: S49051; оценка по BLAST: 530). Это сходство с протеином, участвующим в пути биосинтеза другого близкого антибиотика, в высокой степени позволяет полагать, что ген orf1 кодирует также цитохром Р450. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf1, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 13).
Ген orf2 кодирует протеин, имеющий относительно большое сходство с dTDP-6-дезокси-3,4-кетогексулозоизомеразой Aneurinibacillus thermoaerophilus (Pfoestl A. и др., 2003; номер доступа в банк генов: ААO06351; оценка по BLAST: 118). Это сходство в высокой степени позволяет полагать, что ген orf2 кодирует изомеразу, ответственную за реакцию изомеризации, необходимую в биосинтезе одного из сахаров, присутствующих в молекуле спирамицина, причем этим сахаром может быть микароза (см. фиг.5). Осуществляли инактивацию гена orf2. Показано, что полученный в результате штамм более не продуцирует спирамицинов. Это подтверждает то, что ген orf2 действительно участвует в биосинтезе спирамицинов.
Ген orf3 кодирует протеин, имеющий относительно большое сходство с несколькими аминотрансферазами. В частности, протеин, кодируемый геном orf3, обладает значительным сходством с аминотрансферазой Streptomyces antibioticus (Draeger G. и др., 1999; номер доступа в банк генов: ААF59939; оценка по BLAST: 431). Это сходство с протеином, участвующим в пути биосинтеза другого близкого антибиотика, в высокой степени позволяет полагать, что ген orf3 кодирует 3-аминотрансферазу, ответственную за реакцию трансаминирования, необходимую в биосинтезе одного из аминосахаров спирамицинов (см. фиг.5). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf3, имеет большое сходство с другими протеинами подобной функции в случае других организмов (см. таблицу 15).
Осуществляли инактивацию гена orf3. Таким образом, показано, что полученный в результате штамм не продуцирует больше спирамицинов. Это подтверждает то, что ген orf3 действительно участвует в биосинтезе спирамицинов. Фермент, кодируемый этим геном, следовательно, является в самом деле ответственным за стадию биоконверсии, существенную в биосинтезе спирамицинов. Продуцирование спирамицинов может быть дополнено экспрессией протеина TylB S.fradiae (см. пример 23). Это свидетельствует о том, что ген orf3 кодирует 3-аминотрансферазу, ответственную за реакцию трансаминирования, необходимую в биосинтезе микаминозы (см. фиг.5). Так как микаминоза представляет собой первый сахар, фиксируемый на платенолиде, ожидают, что штамм, прерванный в orf3 (OS49.67), аккумулирует платенолид.
Исследовали медиаторы биосинтеза штамма, прерванного в гене orf3 (см. пример 20). Эти эксперименты позволили показать, что этот штамм продуцирует две формы платенолида: платенолид А и платенолид В, установленная структура этих двух молекул представлена на фиг.36. Этот штамм также продуцирует платенолид А + микароза и платенолид В + микароза (см. пример 20 и фиг.40). Эти соединения включают сахар, но не содержат микаминозы. Кроме того, если их сравнивают со спирамицином (см. фиг.1), то эти соединения включают микарозу вместо микаминозы. Эти результаты согласуются с участием вырабатываемого геном orf3 продукта в биосинтезе микаминозы и его ролью в качестве 3-аминотрансферазы, ответственной за реакцию трансаминирования, необходимую в биосинтезе микаминозы (см. фиг.5). Можно заметить, что специфичность гликозилирования не кажется абсолютной, поскольку обнаруживают молекулы с микарозой, фиксированной в положении, обычно занимаемом микаминозой (см. фиг.40).
Ген orf4 кодирует протеин, имеющий относительно большое сходство с несколькими NDP-глюкозосинтетазами. В частности, протеин, кодируемый геном orf4, обладает значительным сходством с a-D-глюкозо-1-фосфаттимидилилтрансферазой Streptomyces venezuelae (Xue Y. и др., 1998; номер доступа в банк генов: AAC68682; оценка по BLAST: 404). Это сходство с протеином, участвующим в пути биосинтеза другого близкого антибиотика, в высокой степени позволяет полагать, что ген orf4 кодирует также NDP-глюкозосинтетазу, ответственную за синтез NDP-глюкозы, необходимой в биосинтезе трех атипичных сахаров, встроенных в молекулу спирамицина (см. фиг.4, 5 и 6). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf4, имеет большое сходство с другими протеинами подобной функции в случае других организмов (см. таблицу 16).
трансфераза
трансфераза
Ген orf5 кодирует протеин, обладающий относительно большим сходством с несколькими глюкозодегидратазами. В частности, протеин, кодируемый orf5, обладает значительным сходством с dTDP-глюкозо-4,6-дегидратазой Streptomyces tenebrarius (Li T. B. и др., 2001; номер доступа в банк генов: AAG18457; оценка по BLAST: 476). Это сходство с протеином, участвующим в пути биосинтеза другого близкого антибиотика, в высокой степени позволяет полагать, что ген orf5 кодирует также NDP-глюкозодегидратазу, необходимую в биосинтезе трех атипичных сахаров, встроенных в молекулу спирамицина (см. фиг.4, 5 и 6). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf5, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 17).
таза
таза
таза
Ген orf6 кодирует протеин, обладающий относительно большим сходством с несколькими тиоэстеразами. В частности, протеин, кодируемый orf6, обладает значительным сходством с тиоэстеразой Streptomyces avermitilis (Omura S. и др., 2001; номер доступа в банк генов: BAB69315; оценка по BLAST: 234). Это сходство с протеином, участвующим в пути биосинтеза другого близкого антибиотика, в высокой степени позволяет полагать, что ген orf6 кодирует также тиоэстеразу. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf6, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 18).
Ген orf7 кодирует протеин, обладающий относительно большим сходством с несколькими гексозодегидратазами. В частности, протеин, кодируемый orf7, обладает значительным сходством с dNTP-гексозо-2,3-дегидратазой (кодируемой геном TylCVI) Streptomyces fradiae, участвующей в биосинтезе тилозина (Merson-Davies L. A. и др., 1994; номер доступа в банк генов: AAF29379; оценка по BLAST: 461). Это сходство с протеином, участвующим в пути биосинтеза другого близкого антибиотика, в высокой степени позволяет полагать, что ген orf7 кодирует также гексозо-2,3-дегидратазу, необходимую в биосинтезе двух атипических сахаров, встроенных в молекулу спирамицина (см. фиг.4 и 6). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf7, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 19).
Ген orf8 кодирует протеин, обладающий относительно большим сходством с несколькими аминотрансферазами. В частности, протеин, кодируемый orf8, обладает значительным сходством с аминотрансферазой, возможно участвующей в биосинтезе форозамина в случае Saccharopolyspora spinosa (Waldron C. и др., 2001; номер доступа в банк генов: AAG23279; оценка по BLAST: 465). Это сходство с протеином, участвующим в пути биосинтеза другого близкого антибиотика, в высокой степени позволяет полагать, что ген orf8 кодирует 4-аминотрансферазу, ответственную за реакцию трансаминирования, необходимую в биосинтезе форозамина (см. фиг.6). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf8, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 20).
Осуществляли инактивацию гена orf8. Таким образом, показано, что полученный в результате штамм не продуцирует больше спирамицинов. Это подтверждает то, что ген orf8 действительно участвует в биосинтезе спирамицинов. Фермент, кодируемый этим геном, следовательно, является в самом деле ответственным за стадию биоконверсии, существенную в биосинтезе спирамицинов. В пользу признания действительной гипотезы о роли, которую играет вырабатываемый геном orf8 продукт в биосинтезе форозамина, говорит тот факт, что инактивированный мутант гена orf8 продуцирует фороцидин, следовательно, этот мутант блокирован на стадии фороцидина и не продуцирует неоспирамицин (см. фиг.7 и пример 25). Эти результаты согласуются с участием вырабатываемого геном orf8 продукта в биосинтезе форозамина (см. фиг.6).
Ген orf9 уже был идентифицирован в случае Streptomyces ambofaciens был назван srmX согласно Geistlich и др. (Geistlich M. и др., 1992). Сходство протеина, кодируемого этим геном, с несколькими метилтрансферазами, участвующими в пути биосинтеза других близких антибиотиков, в высокой степени позволяет полагать, что ген orf9 кодирует метилтрансферазу, ответственную за реакцию метилирования, необходимую в биосинтезе микаминозы или форозамина (см. фиг.5 и 6). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf9, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 21).
Ген orf10 уже был идентифицирован в случае Streptomyces ambofaciens и назван srmR согласно Geistlich и др. (Geistlich M. и др., 1992). Протеин, кодируемый этим геном, участвует в регуляции пути биосинтеза спирамицинов в случае Streptomyces ambofaciens. Осуществляли инактивацию гена orf10. Таким образом, показано, что полученный в результате штамм не продуцирует больше спирамицинов. Это подтверждает то, что ген orf10 действительно участвует в биосинтезе спирамицинов. Протеин, кодируемый этим геном, следовательно, является в самом деле существенным в биосинтезе спирамицинов.
С другой стороны, был определен сайт инициации трансляции orf10 и показано, что сверхпродуцирование этого гена вызывает повышение продуцирования спирамицинов. Сайт инициации трансляции соответствует ATG, расположенному выше ATG, предложенного Geistlich и др. (Geistlich M. и др., 1992). Кроме того, показано, что этот 5'-конец является существенным для функции orf10, поскольку укороченная на 5' мРНК является неактивной (см. пример 17). Для достижения искомого эффекта в отношении продуцирования спирамицинов, следовательно, существенным является то, чтобы происходило сверхпродуцирование orf10, не остерегаясь экспрессирования укороченной на 5' мРНК гена orf10.
Ген orf11с уже был идентифицирован в случае Streptomyces ambofaciens и назван srmB согласно Geistlich и др. (Geistlich M. и др., 1992) и Schoner и др. (Schoner B. и др., 1992). Протеин, кодируемый этим геном, участвует в резистентности к спирамицину в случае Streptomyces ambofaciens и является переносчиком типа АВС.
Ген orf12 кодирует протеин, обладающий относительно большим сходством с несколькими гексозодегидратазами. В частности, протеин, кодируемый orf12, обладает значительным сходством с NDP-гексозо-3,4-дегидратазой, кодируемой геном UrdQ Streptomyces fradiae и участвующей в биосинтезе урдамицина (Hoffmeister D. и др., 2000; номер доступа в банк генов: AAF72550; оценка по BLAST: 634). Это сходство с протеином, участвующим в пути биосинтеза другого близкого антибиотика, в высокой степени позволяет полагать, что ген orf12 кодирует 3,4-дегидратазу, ответственную за реакцию дегидратации, необходимую в биосинтезе форозамина (см. фиг.6). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf12, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 22).
Осуществляли инактивацию гена orf12. Показано, что полученный в результате штамм не продуцирует больше спирамицинов. Это подтверждает то, что ген orf12 действительно участвует в биосинтезе спирамицинов. Фермент, кодируемый этим геном, следовательно, является в самом деле ответственным за стадию биоконверсии, существенную в биосинтезе спирамицина. В пользу признания действительной гипотезы о роли, которую играет Orf12 в биосинтезе форозамина, говорит тот факт, что инактивированный мутант гена orf12 не продуцирует больше форозамин. Однако, он продуцирует незначительное количество фороцидина. Следовательно, этот мутант блокирован на стадии фороцидина и не продуцирует неоспирамицин (см. фиг.7 и пример 26). Этот мутант, кроме того, продуцирует соединение структуры, представленной на фиг.38. Это последнее соединение включает два сахара, микаминозу и микарозу, но не включает форозамин. Кроме того, если его сравнивают со структурой спирамицина (см. фиг.1), то это соединение включает сахар микарозу вместо ожидаемого форозамина. Эти результаты согласуются с участием вырабатываемого геном orf12 продукта в биосинтезе форозамина (см. фиг.6). Можно заметить, что специфичность гликозилирования не является абсолютной, поскольку обнаруживают молекулы, в которых микароза фиксирована в положении, обычно занимаемом форозамином (см. фиг.38).
Ген orf13c кодирует протеин, обладающий относительно большим сходством с протеином неизвестной функции в случае Streptomyces coelicolor. Этот протеин был назван SC4H2.17 (номер доступа в банк генов: Т35116; оценка по BLAST: 619). Протеин, кодируемый геном orf13c, также обладает большим сходством с другими протеинами в случае других организмов (см. таблицу 23).
Никакая точная функция не была определена у протеинов, близких таковой, кодируемой геном orf13c. Была осуществлена инактивация гена orf13c с целью изучения функции этого гена в пути биосинтеза спирамицинов в случае Streptomyces ambofaciens. Показано, что полученный в результате штамм продуцирует спирамицины. Это указывает на то, что ген orf13c не является существенным в биосинтезе спирамицинов и что он не является существенным для выживания бактерии. Фермент, кодируемый этим геном, следовательно, не является ответственным за стадию биоконверсии, существенную в биосинтезе спирамицинов.
Ген orf14 кодирует протеин, обладающий относительно большим сходством с предполагаемой редуктазой (Redenbach M. и др., 1996; Bentley и др., 2002; номер доступа в банк генов: САВ90862; оценка по BLAST: 147).
Осуществляли инактивацию гена orf14. Показано, что полученный в результате штамм не продуцирует больше спирамицинов. Это подтверждает то, что ген orf14 действительно участвует в биосинтезе спирамицинов. Фермент, кодируемый этим геном, следовательно, является в самом деле ответственным за стадию биоконверсии, существенную в биосинтезе спирамицина. Исследованы медиаторы биосинтеза в случае штамма, прерванного в гене orf14 (см. пример 20). Эти эксперименты позволили показать, что этот штамм продуцирует платенолид А, но не продуцирует платенолид В (см. фиг.36).
Ген orf15c кодирует протеин, обладающий относительно большим сходством с несколькими кеторедуктазами. В частности, протеин, кодируемый orf15c, обладает значительным сходством с 3-кеторедуктазой в случае Streptomyces antibioticus (номер доступа в банк генов: T72550; оценка по BLAST: 285). Это сходство в высокой степени позволяет полагать, что ген orf15c кодирует 3-кеторедуктазу, ответственную за реакцию восстановления, необходимую в биосинтезе форозамина (см. фиг.6). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf15с, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 24).
таза
Ген orf16 кодирует протеин, обладающий относительно большим сходством с несколькими изомеразами. В частности, протеин, кодируемый orf16, обладает значительным сходством с NDP-гексозо-3,4-изомеразой в случае Streptomyces fradiae (Gandecha и др., 1997; номер доступа в банк генов: САА57471; оценка по BLAST: 209). Это сходство в высокой степени позволяет полагать, что ген orf16 кодирует протеин, участвующий в биосинтезе одного из сахаров спирамицина (см. фиг.5 и 6). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf16, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 25).
Ген orf17 кодирует протеин, обладающий относительно большим сходством с несколькими гликозилтрансферазами. В частности, протеин, кодируемый orf17, обладает значительным сходством с гликозилтрансферазой Streptomyces venezuelae (Xue Y. и др., 1998; номер доступа в банк генов: ААC68677; оценка по BLAST: 400). Сходство протеина, кодируемого геном orf17, с несколькими гликозилтрансферазами, участвующими в пути биосинтеза других близких антибиотиков, в высокой степени позволяет полагать, что этот ген кодирует также гликозилтрансферазу. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf17, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 26).
Ген orf18 кодирует протеин, обладающий относительно большим сходством с несколькими гликозилтрансферазами. В частности, протеин, кодируемый orf18, обладает значительным сходством с гликозилтрансферазой Streptomyces rishiriensis (Wang и др., 1998; номер доступа в банк генов: ААG29785; оценка по BLAST: 185). Сходство протеина, кодируемого геном orf18, с несколькими гликозилтрансферазами, участвующими в пути биосинтеза других близких антибиотиков, в высокой степени позволяет полагать, что этот ген также кодирует гликозилтрансферазу. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf18, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 27).
Ген orf19 кодирует протеин, обладающий относительно большим сходством с несколькими кеторедуктазами. В частности, протеин, кодируемый orf19, обладает значительным сходством с NDP-гексозо-4-кеторедуктазой (TylCIV) Streptomyces fradiae (Bate и др., 2000; номер доступа в банк генов: ААD41822; оценка по BLAST: 266). Сходство протеина, кодируемого геном orf19, с этой кеторедуктазой, участвующей в пути биосинтеза близкого антибиотика, в высокой степени позволяет полагать, что этот ген кодирует также 4-кеторедуктазу, ответственную за реакцию восстановления, необходимую в биосинтезе микарозы (см. фиг.4). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf19, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 28).
редуктаза
Ген orf20 кодирует протеин, обладающий относительно большим сходством с несколькими гексозоредуктазами. В частности, протеин, кодируемый orf20, обладает значительным сходством с геном EryBII Saccharopolyspora erythraea, который кодирует dTDP-4-кето-L-6-дезоксигексозо-2,3-редуктазу (Summers R.G. и др., 1997; номер доступа в банк генов: ААB84068; оценка по BLAST: 491). Сходство протеина, кодируемого геном orf20, с несколькими гексозоредуктазами, участвующими в пути биосинтеза других близких антибиотиков, в высокой степени позволяет полагать, что этот ген кодирует 2,3-редуктазу, ответственную за реакцию восстановления, необходимую в биосинтезе микарозы (см. фиг.4). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf20, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 29).
Ген orf21c кодирует протеин, обладающий относительно большим сходством с несколькими гексозометилтрансферазами. В частности, протеин, кодируемый orf21с, обладает значительным сходством с геном TylCIII Streptomyces fradiae, который кодирует NDP-гексозо-3-С-метилтрансферазу (Bate N. и др., 2000; номер доступа в банк генов: ААD41823; оценка по BLAST: 669). Сходство протеина, кодируемого геном orf21c, с несколькими гексозометилтрансферазами, участвующими в пути биосинтеза других близких антибиотиков, в высокой степени позволяет полагать, что этот ген кодирует гексозометилтрансферазу, ответственную за реакцию метилирования, необходимую в биосинтезе микарозы (см. фиг.4). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf21с, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 30).
Ген orf22c кодирует протеин, обладающий относительно большим сходством с протеином, кодируемым геном fkbH Streptomyces hygroscopicus var. ascomyceticus, который кодирует фермент, участвующий в биосинтезе метоксималонила (Wu K. и др., 2000; номер доступа в банк генов: ААF86387; оценка по BLAST: 463). Сходство протеина, кодируемого геном orf22c, с этим протеином, участвующим в пути биосинтеза другого близкого макролида, в высокой степени позволяет полагать, что этот ген кодирует также фермент, участвующий в биосинтезе метоксималонила в случае Streptomyces ambofaciens (см. фиг.8).
Ген orf23c кодирует протеин, обладающий относительно большим сходством с протеином, кодируемым геном fkbI Streptomyces hygroscopicus var. ascomyceticus, который кодирует ацил-СоА-дегидрогеназу, участвующую в биосинтезе метоксималонила (Wu K. и др., 2000; номер доступа в банк генов: ААF86388; оценка по BLAST: 387). Сходство протеина, кодируемого геном orf23c, с несколькими ацил-СоА-дегидрогеназами, участвующими в пути биосинтеза других близких антибиотиков, в высокой степени позволяет полагать, что этот ген кодирует ацил-СоА-дегидрогеназу, участвующую в биосинтезе метоксималонила (см. фиг.8). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf23c, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 31).
Ген orf24c кодирует протеин, обладающий относительно большим сходством с протеином, кодируемым геном fkbJ Streptomyces hygroscopicus var. ascomyceticus, который кодирует протеин связывания ацильной группы (протеин-носитель ацильной группы (АСР)), участвующий в биосинтезе метоксималонила (Wu K. и др., 2000; номер доступа в банк генов: ААF86389; оценка по BLAST: 87). Сходство протеина, кодируемого геном orf24c, с этим протеином, участвующим в пути биосинтеза другого близкого макролида, в высокой степени позволяет полагать, что этот ген кодирует протеин, участвующий в биосинтезе метоксималонила в случае Streptomyces ambofaciens (см. фиг.8).
Ген orf25c кодирует протеин, обладающий относительно большим сходством с протеином, кодируемым геном fkbK Streptomyces hygroscopicus var. ascomyceticus, который кодирует ацил-СоА-дегидрогеназу, участвующую в биосинтезе метоксималонила (Wu K. и др., 2000; номер доступа в банк генов: ААF86390; оценка по BLAST: 268). Сходство протеина, кодируемого геном orf25c, с несколькими ацил-СоА-дегидрогеназами, участвующими в пути биосинтеза других близких антибиотиков, в высокой степени позволяет полагать, что этот ген кодирует ацил-СоА-дегидрогеназу, участвующую в биосинтезе метоксималонила (см. фиг.8). Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf25c, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 32).
Ген orf26 кодирует протеин, обладающий идентичностью 65% (определенной по программе BLAST) с протеином, кодируемым геном tylCV, который кодирует микарозилтрансферазу, участвующую в биосинтезе тилозина в случае Streptomyces fradiae (Bate N. и др., 2000; номер доступа в банк генов: ААD41824; оценка по BLAST: 471). Более конкретно, TylCV представляет собой гликозилтрансферазу, связывающую молекулу микарозы во время синтеза тилозина. Это сходство с протеином, участвующим в пути биосинтеза другого, относительно близкого антибиотика и в особенности в переносе микарозы, позволяет полагать, что ген orf26 кодирует гликозилтрансферазу. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf26, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 33).
Ген orf27 кодирует протеин, обладающий идентичностью 70% (определено по программе BLAST) с протеином, кодируемым геном tylCVII, который кодирует NDP-гексозо-3,5-(или 5-)эпимеразу, участвующую в биосинтезе тилозина в случае Streptomyces fradiae (Bate N. и др., 2000; номер доступа в банк генов: ААD41825; оценка по BLAST: 243). Более конкретно, TylCVII представляет собой гексозо-3,5-(или 5-)эпимеразу, участвующую в биосинтезе микарозы. Это сходство с протеином, участвующим в пути биосинтеза другого, относительно близкого антибиотика и в особенности в биосинтезе микарозы, позволяет полагать, что ген orf27 кодирует эпимеразу. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf27, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 34). Анализ близких последовательностей, получаемых по программе BLAST, в высокой степени позволяет полагать, что ген orf27 кодирует 5-эпимеразу, ответственную за реакцию эпимеризации, необходимую в биосинтезе микарозы (см. фиг.4).
эпимераза
Последовательность orf28c, во-первых, определена частично, поскольку последовательность участка длиной примерно 450 пар оснований была определена только после повторного секвенирования (этот участок отмечен символами “N” в неполной последовательности SEQ ID № 106). Частичная последовательность этой открытой рамки считывания (SEQ ID № 111) тем не менее была использована для анализа с помощью различных информационных программ, как объяснено выше. Таким образом, определено, что ген orf28c кодирует протеин, обладающий идентичностью 64% по определенной последовательности (SEQ ID № 112, которая представляет собой частичную последовательность протеина Orf28c) (определенная по программе BLAST) с протеином, кодируемым геном acyB2, который кодирует протеин-регулятор, участвующий в биосинтезе карбомицина в случае Streptomyces thermotolerans (Arisawa A. и др., 1993; номер доступа в банк генов: JC2032; оценка по BLAST: 329). Это сходство с протеином, участвующим в пути биосинтеза относительно близкого антибиотика, позволяет полагать, что ген orf28c кодирует протеин-регулятор, участвующий в биосинтезе спирамицинов. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf28c, также обладает большим сходством с протеином TylR, который является протеином-регулятором, участвующим в биосинтезе тилозина в случае Streptomyces fradiae (Bate N. и др., 1999; номер доступа в банк генов: AAF29380; оценка по BLAST: 167).
Ген orf28c был амплифицирован благодаря олигонуклеотидам, расположенным с той и другой стороны неопределенной последовательности, и субклонирован в экспрессирующем векторе. Таким образом, показано, что сверхпродуцирование гена orf28c значительно повышает продуцирование спирамицинов штамма OSC2 (см. пример 24). Это показывет, что сверхпродуцирование гена orf28c приводит к повышению продуцирования спирамицинов и подтверждает его роль в качестве регулятора пути биосинтеза спирамицинов.
Частичная последовательность orf28c, следовательно, была дополнена и была определена недостающая область длиной около 450 пар оснований (см. SEQ ID № 140 и SEQ ID № 141). Полная последовательность этой открытой рамки считывания (SEQ ID № 141) была использована для анализа с помощью различных информационных программ, как объяснено выше. Таким образом, определено, что ген orf28c кодирует протеин, обладающий идентичностью 69% по определенной последовательности (SEQ ID № 142, которая представляет собой полную последовательность протеина Orf28c) (определенная по программе BLAST) с протеином, кодируемым геном acyB2, который кодирует протеин-регулятор, участвующий в биосинтезе карбомицина в случае Streptomyces thermotolerans (Arisawa A. и др., 1993; номер доступа в банк генов: JC2032; оценка по BLAST: 451). Это сходство с протеином, участвующим в регуляции биосинтеза относительно близкого антибиотика, позволяет полагать, что ген orf28c кодирует протеин-регулятор, участвующий в биосинтезе спирамицинов. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf28c, также обладает большим сходством с протеином TylR, который является протеином-регулятором, участвующим в регуляции биосинтеза тилозина в случае Streptomyces fradiae (Bate N. и др., 1999; номер доступа в банк генов: AAF29380; оценка по BLAST: 224). Результаты сверхпродуцирования этого гена (см. пример 24) подтверждают его роль в качестве регулятора пути биосинтеза спирамицинов.
Была осуществлена инактивация гена orf28c. Таким образом, показано, что полученный в результате штамм не продуцирует больше спирамицинов. Это подтверждает то, что ген orf28c действительно участвует в биосинтезе спирамицинов и является существенным в биосинтезе спирамицинов. Эти результаты вместе с результатами по сверхпродуцированию этого гена (см. пример 24) согласуются с ролью активатора, существенного в биосинтезе спирамицинов Orf28c.
Ген orf29 кодирует протеин, обладающий идентичностью 31% (определено по программе BLAST) с вероятной гликозилгидролазой, локализованной в группе генов, участвующей в биосинтезе сорафена А (противогрибковое средство класса поликетидов) в случае Sorangium cellulosum (Ligon J. и др., 2002; номер доступа в банк генов: ААK19890; оценка по BLAST: 139). Это сходство с протеином, участвующим в пути биосинтеза относительно близкой молекулы, позволяет полагать, что ген orf29 кодирует протеин, обладающий активностью гликозилгидролазы. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf29, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. табл.35). Анализ последовательности протеина, кодируемого orf29, по программе CD-search (см. выше) также позволяет полагать, что ген orf29 кодирует гликозилгидролазу.
Анализ протеиновой последовательности, следующей из orf29, по программе SignalP (http://www.cbs.dtu.dk/services/SignalP/) (Nielsen H. и др., 1997) показывает, что этот протеин имеет С-концевую сигнальную последовательность с предсказанным сайтом расщепления между положениями 30 и 31 (QSA/QA). Можно предсказать, что этот протеин является внеклеточным. Он может, также как гликозилгидролаза, играть роль в реактивации инактивированного спирамицина путем гликозилирования с помощью гликозилтрансфераз GimA и/или GimB (Gourmelen и др., 1998).
Ген orf30c кодирует протеин, обладающий идентичностью 31% (определено по программе BLAST) с сахариднуклеозиддифосфатэпимеразой Corynebacterium glutamicum (номер доступа в банк генов: NP_600590; оценка по BLAST: 89). Это сходство позволяет полагать, что ген orf30c кодирует эпимеразу. Эта гипотеза подкрепляется тем фактом, что анализ последовательности по программе CD-search (см. выше) позволяет полагать, что ген orf30c кодирует эпимеразу.
Ген orf30c имеет два возможных кодона инициаций (см. SEQ ID № 115), которые дают два возможных протеина длиной, соответственно, 345 аминокислот и 282 аминокислоты (SEQ ID № 116 и SEQ ID № 117). Однако использование кодонов является типичным для Streptomyces только исходя из второго ATG, к тому же протеиновая последовательность, следующая из последовательности между первым ATG и вторым ATG, не сопоставима путем выравнивания с идентифицированными близкими последовательностями, тогда как наиболее короткая протеиновая последовательность (исходя из второго ATG: SEQ ID № 144) хорошо сопоставима путем выравнивания с началом этих протеинов. Следовательно, можно сделать вывод, что второй ATG представляет собой хороший кодон инициации и что последовательность этой открытой рамки считывания (orf), следовательно, является таковой, представленной в виде SEQ ID № 143, которая, один раз выраженная, соответствует протеину, соответствующему последовательности SEQ ID № 144.
Ген orf31 кодирует протеин, обладающий идентичностью 52% (определено по программе BLAST) с оксидоредуктазой в Streptomyces coelicolor (номер доступа в банк генов: NP_631148; оценка по BLAST: 261). Это сходство позволяет полагать, что ген orf31 кодирует редуктазу. Эта гипотеза подкрепляется тем фактом, что анализ последовательности по программе CD-search (см. выше) позволяет также полагать, что ген orf31 кодирует редуктазу. Эта гипотеза также подкрепляется тем фактом, что протеин, кодируемый геном orf31, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 36).
Была осуществлена инактивация гена orf31. Таким образом, показано, что полученный в результате штамм не продуцирует больше спирамицинов. Это подтверждает то, что ген orf31 действительно участвует в биосинтезе спирамицинов. Фермент, кодируемый этим геном, следовательно, является в самом деле ответственным за стадию биоконверсии, существенную в биосинтезе спирамицинов.
Последовательность orf32c сначала была определена частично (см. пример 19), поскольку кодирующая последовательность в 5' была определена только потом. Частичная последовательность этой открытой рамки считывания (SEQ ID № 120) тем не менее была использована для анализа с помощью различных информационных программ, как объяснено выше. Таким образом, определено, что ген orf32c кодирует протеин, обладающий идентичностью 47% по определенной последовательности (SEQ ID № 121, которая представляет собой частичную последовательность протеина Orf32c) (определено по программе BLAST) с протеином-регулятором семейства GntR в случае Streptomyces coelicolor (номер доступа в банк генов: NP_625576; оценка по BLAST: 229). Это сходство позволяет полагать, что ген orf32c кодирует регулятор транскрипции семейства GntR. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf32c, обладает большим сходством с другими протеинами подобной функции в случае других организмов.
Частичная последовательность orf32c затем была дополнена и была определена недостающая область (см. SEQ ID № 140 и SEQ ID № 145). Полная последовательность этой открытой рамки считывания кодирует протеин, обладающий идентичностью 44% (определено по программе BLAST) с протеином-регулятором семейства GntR в случае Streptomyces avermitilis (номер доступа в банк генов: NP_824604; оценка по BLAST: 282). Это сходство позволяет полагать, что ген orf32c кодирует регулятор транскрипции семейства GntR. Эта гипотеза подкрепляется тем фактом, что протеин, кодируемый геном orf32c, обладает большим сходством с другими протеинами подобной функции в случае других организмов (см. таблицу 37).
Осуществляли инактивацию гена orf32c с целью изучения функции этого гена в пути биосинтеза спирамицинов в случае Streptomyces ambofaciens. Показано, что полученный в результате штамм продуцирует спирамицины. Это указывает на то, что ген orf32c не является существенным в биосинтезе спирамицинов и что он не является существенным для выживания бактерии.
Ген orf33 кодирует протеин, обладающий идентичностью 49% (определено по программе BLAST) с гипотетическим протеином Xanthomnas campestris (номер доступа в банк генов: NP_635564; оценка по BLAST: 54).
Последовательность orf34c является частичной. В самом деле, сравнения, осуществленные между продуктом этой открытой рамки считывания и банками данных, позволяют полагать, что С-концевая часть этого протеина отсутствует в продукте, следующем из нуклеотидной последовательности, и, следовательно, что эта открытая рамка считывания является более длинной и продолжается вне секвенированной области. Частичная последовательность этой ORF, тем не менее, была использована для анализа с помощью различных информационных программ, как указанные выше. Таким образом, определено, что ген orf34c кодирует протеин, обладающий идентичностью 91% по определенной последовательности (SEQ ID № 150, которая представляет собой частичную последовательность протеина Orf34c) (определено по программе BLAST) с арабинофуранозидазой в случае Streptomyces coelicolor (Bentley и др., 2002; номер доступа в банк генов: NP_630049; оценка по BLAST: 654). В случае S. coelicolor, ген, кодирующий эту арабинофуранозидазу, по-видимому, не участвует в биосинтезе вторичного метаболита. Этот ген в S. ambofaciens, следовательно, вероятно не участвует в биосинтезе спирамицина.
Объектом настоящего изобретения являются также полинуклеотиды, гибридизующиеся в условиях гибридизации высокой жесткости по меньшей мере с одним из полинуклеотидов, соответствующих последовательности SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 107, 109, 111, 113, 115, 118, 120, 141, 143, 145, 147 и 149 или с одним из его вариантов или с одной из последовательностей, происходящих от них в соответствии с вырожденностью генетического кода. Предпочтительно, эти указанные полинуклеотиды выделяли из бактерии рода Streptomyces, более предпочтительно, эти полинуклеотиды кодируют протеины, участвующие в биосинтезе макролида, и, еще более предпочтительно, эти полинуклеотиды кодируют протеин, обладающий активностью, подобной протеину, кодируемому полинуклеотидами, с которыми они гибридизуются. Условия гибридизации высокой жесткости могут быть определены как условия гибридизации, неблагоприятствующие гибридизации негомологичных нитей нуклеиновых кислот. Условиями гибридизации высокой жесткости могут быть, например, условия, описанные как условия гибридизации в буфере, описанном Church и Gilbert (Church & Gilbert, 1984), при температуре от 55°С до 65°С, причем, предпочтительно, температура гибридизации составляет 55°С, еще более предпочтительно, температура гибридизации составляет 60°С и, в высшей степени предпочтительно, температура гибридизации составляет 65°С, с последующими одной или несколькими промывками, осуществляемыми в буфере 2Х SSC при температуре от 55°С до 65°С, причем, предпочтительно, эта температура составляет 55°С, еще более предпочтительно, эта температура составляет 60°С и, в высшей степени предпочтительно, эта температура составляет 65°С, с последующими одной или несколькими промывками, осуществляемыми в буфере 0,5Х SSC при температуре от 55°С до 65°С, причем, предпочтительно, эта температура составляет 55°С, еще более предпочтительно, эта температура составляет 60°С и, в высшей степени предпочтительно, эта температура составляет 65°С. Вышеописанные условия гибридизации могут быть адаптированы в зависимости от длины нуклеиновой кислоты, гибридизацию которой исследуют, или типа выбранной маркировки, согласно способам, известным специалисту в данной области. Пригодные условия гибридизации, например, могут быть адаптированы согласно работе F. Ausubel и др., 2002.
Изобретение относится также к полинуклеотиду, обладающему по меньшей мере 70%, более предпочтительно, 80%, более предпочтительно, 85%, еще более предпочтительно, 90%, еще более предпочтительно, 95% и, в высшей степени предпочтительно, 98% идентичности, по нуклеотидам, с полинуклеотидом, включающим по меньшей мере 10, 12, 15, 18, 20-25, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 350, 400, 450, 500, 550, 600, 650, 700, 750, 800, 850, 900, 950, 1000, 1050, 1100, 1150, 1200, 1250, 1300, 1350, 1400, 1450, 1500, 1550, 1600, 1650, 1700, 1750, 1800, 1850 или 1900 последовательных нуклеотидов полинуклеотида, выбираемого из группы, состоящей из нуклеотидных последовательностей SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 107, 109, 111, 113, 115, 118, 120, 141, 143, 145, 147 и 149 или одного из его вариантов или одной из последовательностей, происходящих от них в соответствии с вырожденностью генетического кода, или полинуклеотида с комплементарной последовательностью. Предпочтительно, эти указанные полинуклеотиды выделяют из бактерии рода Streptomyces, более предпочтительно, эти полинуклеотиды кодируют протеины, участвующие в биосинтезе макролида, и, еще более предпочтительно, эти полинуклеотиды кодируют протеины, обладающие активностями, подобными протеинам, кодируемым полинуклеотидами, с которыми они обладают идентичностью. В высшей степени предпочтительно, полинуклеотид согласно изобретению выбирают из группы, состоящей из нуклеотидных последовательностей SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 107, 109, 111, 113, 115, 118, 120, 141, 143, 145, 147 и 149 или полинуклеотида с комплементарной последовательностью.
Оптимальное сопоставление путем выравнивания последовательностей для сравнения может быть осуществлено компьютерным способом с помощью известных алгоритмов, например, таковых набора программ FASTA (Pearson W.R. & Lipman D.J., 1988; и Pearson W.R., 1990), доступных, в частности, из центра ресурсов INFOBIOGEN, Evry, Франция. В качестве пояснения, процент идентичности последовательности может быть определен с помощью пакета программ LFASTA (Chao K.-M. и др., 1992) или LALIGN (Huang X. и Miller W., 1991). Программы LFASTA и LALIGN составляют часть пакета программ FASTA. LALIGN направлена на оптимальные локальные сопоставления путем выравнивания: эта программа более точная, однако, также более медленная, чем LFASTA.
Другой аспект изобретения относится к полипептиду, получаемому в результате экспрессии нуклеотидной последовательности, такой, как указанная выше. Предпочтительно, полипептиды согласно изобретению обладают по меньшей мере 70%, более предпочтительно, 80%, более предпочтительно, 85%, еще более предпочтительно, 90%, еще более предпочтительно, 95% и, в высшей степени предпочтительно, 98% идентичности, по аминокислотам, с полипептидом, включающим по меньшей мере 10, 15, 20, 30-40, 50, 60, 70, 80, 90,100, 120, 140, 160, 180, 200, 220, 240, 260, 280, 300, 320, 340, 360, 380, 400, 420, 440, 460, 480, 500, 520, 540, 560, 580, 600, 620 или 640 последовательных аминокислот полипептида, выбираемого среди SEQ ID № 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 27, 29, 31, 32, 33, 35, 37, 38, 39, 41, 42, 44, 46, 48, 50, 51, 52, 54, 55, 56, 57, 58, 59, 61, 63, 65, 67, 69, 71, 73, 75, 77, 79, 81, 83, 85, 108, 110, 112, 114, 116, 117, 119, 121, 142, 144, 146, 148 и 150 или одной из этих последовательностей, если не считать того, что, в целом, на протяжении вышеуказанной последовательности одна или несколько аминокислот заменены, встроены или подвергнуты делеции без изменения функциональных свойств, или одного из вариантов этих последовательностей. Предпочтительно, полипептиды согласно изобретению экспрессируются в нативном состоянии бактерией рода Streptomyces, более предпочтительно, эти полипептиды участвуют в биосинтезе макролида и, еще более предпочтительно, эти полипептиды обладают активностью, подобной таковой полипептида, с которым он обладает идентичностью. Предпочтительно, полипептид согласно изобретению выбирают из группы, состоящей из полипептидных последовательностей SEQ ID № 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 27, 29, 31, 32, 33, 35, 37, 38, 39, 41, 42, 44, 46, 48, 50, 51, 52, 54, 55, 56, 57, 58, 59, 61, 63, 65, 67, 69, 71, 73, 75, 77, 79, 81, 83, 85, 108, 110, 112, 114, 116, 117, 119, 121, 142, 144, 146, 148 и 150 или одной из этих последовательностей, если не считать того, что, в целом, по всей длине вышеуказанной последовательности одна или несколько аминокислот заменены, встроены или подвергнуты делеции без изменения функциональных свойств, или одного из вариантов этих последовательностей. В высшей степени предпочтительно, полипептид согласно изобретению выбирают из группы, состоящей из полипептидных последовательностей SEQ ID № 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 27, 29, 31, 32, 33, 35, 37, 38, 39, 41, 42, 44, 46, 48, 50, 51, 52, 54, 55, 56, 57, 58, 59, 61, 63, 65, 67, 69, 71, 73, 75, 77, 79, 81, 83, 85, 108, 110, 112, 114, 116, 117, 119, 121, 142, 144, 146, 148 и 150.
Оптимальное сопоставление путем выравнивания последовательностей для сравнения может быть осуществлено компьютерным способом с помощью известных алгоритмов, например, таковых набора программ FASTA (Pearson W.R. & Lipman D.J., 1988; и Pearson W.R., 1990), доступных, в частности, из центра ресурсов INFOBIOGEN, Evry, Франция. В качестве пояснения, процент идентичности последовательности может быть определен с помощью пакета программ LFASTA (Chao K.-M. и др., 1992) или LALIGN (Huang X. и Miller W., 1991), используя параметры по дефекту, такому, как определен центром ресурсов INFOBIOGEN, Evry, Франция. Программы LFASTA и LALIGN составляют часть пакета программ FASTA. LALIGN направлена на оптимальные локальные сопоставления путем выравнивания: эта программа более точная, однако, также более медленная, чем LFASTA.
Следующий аспект изобретения относится к рекомбинантной ДНК, включающей по меньшей мере один полинуклеотид, такой, как описанный выше. Предпочтительно, эта рекомбинантная ДНК является вектором. Еще более предпочтительно, вектор выбирают из бактериофагов, плазмид, фагемид, интегрирующих векторов, фосмид, космид, шаттл-векторов, ВАС (бактериальная искусственная хромосома) или РАС (Р1-производная искусственная хромосома). В качестве пояснения, в качестве бактериофагов можно назвать фаг лямбда и фаг M13. В качестве плазмид можно назвать плазмиды, реплицирующиеся в E.coli, например, pBR322 и ее производные, pUC18 и ее производные, pUC19 и ее производные, pGB2 и ее производные (Churchward G. и др., 1984), pACYC177 (номер доступа в банк генов: Х06402) и ее производные, pACYC184 (номер доступа в банк генов: Х06403) и ее производные. Можно также назвать плазмиды, реплицирующиеся в Streptomyces, как, например, pIJ101 и ее производные, pSG5 и ее производные, SLP1 и ее производные, SCP2* и ее производные (Kieser и др., 2000). В качестве фагемид можно назвать, в качестве пояснения, pBluescript II и ее производные (выпускаются, в частности, фирмой Stratagene (LaJolla, Калифорния, США)), pGEM-T и ее производные (выпускается фирмой Promega (Мэдисон, Висконсин, США)), λZAPII и ее производные (выпускаются, в частности, фирмой Stratagene (LaJolla, Калифорния, США)). В качестве интегрирующих векторов можно назвать, в качестве пояснения, интегрирующие векторы в случае Streptomyces, как, например, таковые, происходящие от SLP1 (Kieser и др., 2000), таковые, происходящие от pSAM2 (Kieser и др., 2000), векторы, использующие системы интеграции фагов PhiC31 (Kieser и др., 2000) (например, pSET152 (Bierman и др., 1992)) или VWB (Van Mellaert L. и др., 1998), а также векторы, использующие систему интеграции IS117 (Kieser и др., 2000). В качестве фосмид можно назвать, в качестве пояснения, фосмиду pFOS1 (выпускается фирмой New England Biolabs Inc., Beverly, Massachussetts, США) и ее производные. В качестве космид можно назвать, в качестве пояснения, космиду SuperCos и ее производные (выпускаются, в частности, фирмой Stratagene (LaJolla, Калифорния, США)), космиду pWED15 (Wahl и др., 1987) и ее производные. В качестве шаттл-векторов можно назвать, в качестве пояснения, шаттл-плазмиды E.coli/Streptomyces, как, например, pIJ903 и ее производные, ряд плазмид pUWL, pCAO106, pWHM3, pOJ446 и их производные (Kieser и др., 2000), челночные ВАС E.coli/Streptomyces, как, например, таковые, описанные в Международной заявке на патент WO-01/40497. В качестве ВАС (бактериальная искусственная хромосома) можно назвать, в качестве пояснения, ВАС pBeloBAC11 (номер доступа в банк генов: U51113). В качестве РАС (Р1-производная искусственная хромосома) можно назвать, в качестве пояснения, вектор pCYPAC6 (номер доступа в банк генов: AF133437). В высшей степени предпочтительно, вектор, согласно изобретению, выбирают среди pOS49.1, pOS49.11, pOSC49.12, pOS49.14, pOS49.16, pOS49.28, pOS44.1, pOS44.2, pOS44.4, pSPM5, pSPM7, pOS49.67, pOS49.88, pOS49.106, pOS49.120, pOS49.107, pOS49.32, pOS49.43, pOS49.44, pOS49.50, pOS49.99, pSPM17, pSPM21, pSPM502, pSPM504, pSPM507, pSPM508, pSPM509, pSPM1, pBXL1111, pBXL1112, pBXL1113, pSPM520, pSPM521, pSPM522, pSPM523, pSPM524, pSPM525, pSPM527, pSPM528, pSPM34, pSPM35, pSPM36, pSPM37, pSPM38, pSPM39, pSPM40, pSPM41, pSPM42, pSPM43, pSPM44, pSPM45, pSPM47, pSPM48, pSPM50, pSPM51, pSPM52, pSPM53, pSPM55, pSPM56, pSPM58, pSPM72, pSPM73, pSPM515, pSPM519, pSPM74, pSPM75, pSPM79, pSPM83, pSPM107, pSPM543 и pSPM106.
Другой аспект изобретения относится к экспрессионной системе, включающей соответствующий экспрессирующий вектор и клетку-хозяина, позволяющей осуществлять экспрессию одного или нескольких полипептидов, таких как описанные выше, в биологической системе. Экспрессионные векторы согласно изобретению включают нуклеотидную последовательность, кодирующую один или несколько полипептидов, таких как описанные выше, и могут быть предназначены для экспрессии различных полипептидов согласно изобретению в различных клетках-хозяевах, хорошо известных специалистам в данной области. В качестве примера, можно назвать прокариотные экспрессионные системы, такие как экспрессионные системы в случае бактерии E.coli, эукариотные экспрессионные системы, такие как бакуловирусная экспрессионная система, позволяющая осуществлять экспрессию в клетках насекомых, экспрессионные системы, позволяющие осуществлять экспрессию в дрожжевых клетках, экспрессионные системы, позволяющие осуществлять экспрессию в клетках млекопитающих, в частности, в человеческих клетках. Экспрессионные векторы, используемые в таких системах, хорошо известны специалистам в данной области; в том, что касается прокариотных клеток, можно назвать, в качестве пояснения, экспрессионные векторы в случае E. coli, например, семейства рЕТ, выпускаемые фирмой Stratagene (LaJolla, Калифорния, США), векторы семейства GATEWAY, выпускаемые фирмой Invitrogen (Carlsbad, Калифорния, США), векторы семейства pBAD, выпускаемые фирмой Invitrogen (Carlsbad, Калифорния, США), векторы семейства pMAL, выпускаемые фирмой New England Biolabs Inc. (Beverly, Massachussetts, США), экспрессионные векторы, индуктируемые рамнозой, указанные в публикации Wilms B. и др., 2001, и их производные, можно также назвать экспрессионные векторы в Streptomyces, как, например, векторы pIJ4123, pIJ6021, pPM927, pANT849, pANT850, pANT851, pANT1200, pANT1201, pANT1202, и их производные (Kaiser и др., 2000). В том, что касается дрожжевых клеток, можно назвать, в качестве пояснения, вектор pESC, выпускаемый фирмой Stratagene (LaJolla, Калифорния, США). В том, что касается бакуловирусной экспрессионной системы, позволяющей осуществлять экспрессию в клетках насекомых, можно назвать, в качестве пояснения, вектор ВасРАК6, выпускаемый фирмой BD Biosciences Clontech (Palo Alto, Калифорния, США). В том, что касается клеток млекопитающих, можно назвать, в качестве примера, векторы, включающие промотор ранних генов вируса CMV (цитомегаловирус) (например, вектор pCMV и его производные выпускаются фирмой Stratagene (LaJolla, Калифорния, США), или промотор предранних генов (ранний промотор SV40) вакуолизирующего обезьяннего вируса SV40 (например, вектор pSG5, выпускаемый фирмой Stratagene (LaJolla, Калифорния, США).
Изобретение также относится к способу продуцирования полипептида, такого как описанный выше, причем вышеуказанный способ включает следующие стадии:
а) инсерция нуклеиновой кислоты, кодирующей вышеуказанный полипептид, в соответствующий вектор;
b) культивирование, на соответствующей культуральной среде, клетки-хозяина, предварительно трансформированной или трансфектированной с помощью вектора со стадии а);
с) рекуперация кондиционированной культуральной среды или клеточного экстракта, например, путем обработки ультразвуком или за счет осмотического шока;
d) выделение и очистка из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ого) на стадии с), вышеуказанного полипептида;
е) в желательном случае, охарактеризовывание продуцированного рекомбинантного полипептида.
Рекомбинантный полипептид согласно изобретению может быть очищен путем пропускания через соответствующий ряд хроматографических колонок согласно способам, известным специалистам в данной области и описанным, например, F. Ausubel и др. (2002). В качестве примера можно назвать способ с гистидиновой меткой (“Tag-Histidine”), который состоит в добавлении короткой полигистидиновой последовательности к продуцируемому полипептиду, причем этот последний затем может быть очищен на никелевой колонке. Полипептид согласно изобретению также может быть получен способами синтеза in vitro. В качестве пояснения таких способов, полипептид согласно изобретению может быть получен с помощью системы, называемой как “система быстрой трансляции (RTS)”, выпускаемой, в частности, фирмой Roche Diagnostics France S.A., Meylan, Франция.
Следующий аспект изобретения относится к клеткам-хозяевам, в которые введен по меньшей мере один полинуклеотид и/или по меньшей мере одна рекомбинантная ДНК и/или по меньшей мере один экспрессирующий вектор согласно изобретению.
Другой аспект изобретения относится к микроорганизмам, блокированным на одной стадии пути биосинтеза по меньшей мере одного макролида. Представляет интерес, с одной стороны, изучение функции мутированных протеинов и, с другой стороны, реализация микроорганизмов, продуцирующих медиаторы биосинтеза. Эти медиаторы могут быть модифицированы, в случае необходимости, после выделения, либо за счет добавки особых компонентов в среды для продуцирования, либо за счет введения в таким образом мутированные микроорганизмы других генов, кодирующих протеины, которые способны модифицировать медиатор, служащий для них в качестве субстрата. Таким образом, эти медиаторы могут быть модифицированы химическим, биохимическим, ферментативным и/или микробиологическим способом. Микроорганизмы, блокированные на одной стадии пути биосинтеза макролидов, могут быть получены путем инактивации функции одного или нескольких протеинов, участвующих в биосинтезе этого или этих макролидов, в микроорганизмах, являющихся продуцентами этого или этих макролидов. Соответственно инактивированному протеину или инактивированным протеинам, таким образом, можно получать микроорганизмы, блокированные на разных стадиях пути биосинтеза этого или этих макролидов. Инактивация этого или этих протеинов может быть осуществлена любым способом, известным специалистам в данной области, например, путем мутагенеза в гене или генах, кодирующих вышеуказанный или вышеуказанные протеины, или путем экспрессии одной или нескольких антисмысловых РНК, комплементарных РНК или матричных РНК, кодирующих вышеуказанный или вышеуказанные протеины. Мутагенез может быть осуществлен, например, путем облучения, путем воздействия мутагенного химического агента, путем направленного мутагенеза, путем замены гена или любым другим способом, известным специалистам в данной области. Условия, пригодные для такого мутагенеза, могут быть адаптированы, например, согласно указаниям, содержащимся в работах Kieser T. и др. (2000) и Ausubel и др. (2002). Мутагенез может быть осуществлен in vitro или in situ путем супрессии, замены, делеции и/или инсерции одного или нескольких оснований в случае рассматриваемого гена или путем генной инактивации. Этот мутагенез может быть осуществлен в гене, включающем последовательность, такую как описанная выше. Предпочтительно, микроорганизмы, блокированные на одной стадии пути биосинтеза макролидов, представляют собой бактерии рода Streptomyces. Более предпочтительно, инактивацию функции одного или нескольких протеинов, участвующих в биосинтезе рассматриваемого макролида или рассматриваемых макролидов, осуществляют путем мутагенеза. Еще более предпочтительно, рассматриваемым макролидом является спирамицин и микроорганизмами, в которых осуществляют мутагенез или мутагенезы, являются штаммы S. ambofaciens. Более предпочтительно, мутагенез осуществляют в одном или нескольких генах, включающих одну из последовательностей, соответствующих одной или нескольким последовательностям SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 107, 109, 111, 113, 115, 118, 120, 141, 143, 145, 147 и 149.
Предпочтительно, мутагенез или мутагенезы осуществляют путем генной инактивации. В высшей степени предпочтительно, мутагенез состоит в генной инактивации гена, включающего последовательность, соответствующую последовательности SEQ ID № 13.
В качестве примера таких микроорганизмов могут быть названы следующие микроорганизмы: OS49.16 (orf3:; Ωhyg, см. пример 2); OS49.67 (делеция всей orf3, см. пример 6); OS49.107 (orf8:; Ωhyg, см. пример 7); OS49.50 (orf10:; Ωhyg, см. пример 8); SPM21 (orf2::att3Ωaac-, см. пример 10); SPM22 (делеция всей orf2::att3, см. пример 10); SPM501 (orf6*::att1Ωhyg+, см. пример 14); SPM502 (делеция всей orf6*::att1, см. пример 14); SPM507 (orf2::att3Ωaac-, см. пример 11); SPM508 (orf3c::att3Ωaac-, см. пример 12); SPM509 (orf14::att3Ωaac-, см. пример 13); SPM107 (orf28c::att3aac+, см. пример 29); SPM21 (orf31::att3aac+, см. пример 30); SPM543 (orf31::att3aac+, см. пример 30); SPM106 (orf32c::att3aac+, см. пример 31).
Следующий аспект изобретения относится к способу получения медиатора биосинтеза макролида при использовании микроорганизмов, блокированных на одной стадии пути биосинтеза макролидов, таких как описанные выше. Способ состоит в культивировании, на соответствующей культуральной среде, микроорганизма, блокированного на одной стадии пути биосинтеза макролидов, таких как описанные выше, рекуперации кондиционированной культуральной среды или клеточного экстракта, например, путем обработки ультразвуком или осмотического шока, выделении и очистке из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ого) на предыдущей стадии, вышеуказанного медиатора биосинтеза. Условия культивирования таких микроорганизмов могут быть определены согласно способам, хорошо известным специалистам в данной области. Культуральной средой, например, может быть среда МР5 или среда SL11 для Streptomyces и особенно для Streptomyces ambofaciens (Pernodet и др., 1993). Специалист может обратиться, в частности, к работе Kieser и др. (2000) в том, что касается культивирования Streptomyces. Продуцированные медиаторы могут быть рекуперированы любыми, известными специалистам способами. Специалист может обратиться, например, к способам, указанным в патенте США 3000785, и в особенности к способам экстракии спирамицинов, описанным в этом патенте.
Другой объект изобретения относится к способу получения молекулы, происходящей от макролида, при использовании микроорганизмов, блокированных на одной стадии пути биосинтеза макролида, таких как описанные выше. Способ состоит в получении медиатора биосинтеза по описанному выше способу и в модификации таким образом полученного медиатора, в случае необходимости, после выделения из культуральной среды. Условия культивирования таких микроорганизмов могут быть определены согласно хорошо известным специалистам в данной области способам. Культуральной средой, например, может быть среда МР5 или среда SL11 для Streptomyces и особенно для Streptomyces ambofaciens (Pernodet и др., 1993). Специалист может обратиться, в частности, к работе Kieser и др. (2000) в том, что касается культивирования Streptomyces. Продуцированные медиаторы могут быть модифицированы, в случае необходимости, после выделения, либо за счет добавки соответствующих компонентов в среды для продуцирования, либо за счет введения в микроорганизмы других генов, кодирующих протеины, которые способны модифицировать медиатор, служащий для них в качестве субстрата. Эти медиаторы, таким образом, могут быть модифицированы химическим, биохимическим, ферментативным и/или микробиологическим способом. Более предпочтительно, рассматриваемым макролидом является спирамицин и микроорганизмами, в которых осуществляют мутагенез или мутагенезы, являются штаммы S. ambofaciens.
Изобретение также относится к микроорганизму, продуцирующему спирамицин I, но не продуцирующему спирамицины II и III. Этот микроорганизм включает совокупность генов, необходимых в биосинтезе спирамицина I, однако, не продуцирует спирамицины II и III, так как ген, включающий последовательность, соответствующую SEQ ID № 13, или один из ее вариантов или одну из происходящих от них последовательностей в соответствии с вырожденностью генетического кода, и кодирующий полипептид, соответствующий последовательности SEQ ID № 14 или одному из ее варинатов, не экспрессирован или инактивирован. Инактивация этого протеина может быть осуществлена любым способом, известным специалистам в данной области, например, путем мутагенеза в гене, кодирующем вышеуказанный протеин, или путем экспрессии антисмысловой РНК, комплементарной матричной РНК, кодирующей вышеуказанный протеин, имея в виду, что, если экспрессия orf5* изменяется за счет этой манипуляции, необходимо осуществлять другую модификацию, в случае которой корректно экспрессируется ген orf5*. Мутагенез может быть осуществлен в кодирующей последовательности или в некодирующей последовательности, чтобы инактивировать кодируемый протеин или чтобы предотвращать его экспрессию или его трансляцию. Мутагенез может быть осуществлен путем направленного мутагенеза, путем замены гена или любым другим способом, известным специалисту. Пригодные для такого мутагенеза условия, например, могут быть адаптированы согласно указаниям, содержащимся в работах Kieser Т. и др. (2000) и Ausubel и др. (2002). Мутагенез может быть осуществлен in vitro или in situ, путем супрессии, замены, делеции и/или инсерции одного или нескольких оснований в случае рассматриваемого гена или путем генной инактивации. Микроорганизм также может быть получен путем экспрессии генов пути биосинтеза спирамицина, так что они не включают ген, включающий последовательность SEQ ID № 13 или один из ее вариантов или одну из происходящих от них последовательностей в соответствии с вырожденностью генетического кода, и кодирующий полипептид, соответствующий последовательности SEQ ID № 14 или одному из ее вариантов. Предпочтительно, микроорганизмом является бактерия рода Strepromyces. Более предпочтительно, микроорганизм, продуцирующий спирамицин I, но не продуцирующий спирамицины II и III, получают из исходного микроорганизма, продуцирующего спирамицины I, II и III. Еще более предпочтительно, микроорганизм получают путем мутагенеза в гене, включающем последовательность, соответствующую SEQ ID № 13, или один из ее вариантов, или одну из последовательностей, происходящих от них в соответствии с вырожденностью генетического кода, и кодирующем полипептид, соответствующий последовательности SEQ ID № 14 или одному из ее вариантов, имеющему такую же функцию. Еще более предпочтительно, этот мутагенез осуществляют путем генной инактивации. Микроорганизм предпочтительно получают из штамма S. ambociens, продуцирующего спирамицины I, II и III, в котором осуществляют генную инактивацию гена, включающего последовательность, соответствующую SEQ ID № 13, или одну из последовательностей, происходящих от нее в соответствии с вырожденностью генетического кода. В высшей степени предпочтительно, генную инактивацию осуществляют путем делеции полноразмерного гена или части гена, включающего последовательность, соответствующую SEQ ID № 13, или одну из последовательностей, происходящих от нее в соответствии с вырожденностью генетического кода. В качестве примера, штамм SPM502 (orf6*::att1, см. пример 14) может быть указан как микроорганизм, продуцирующий спирамицин I, но не продуцирующий спирамицинов II и III.
Изобретение также относится к микроорганизму, продуцирующему спирамицин I, но не продуцирующему спирамицинов II и III, такому, как описанный выше, отличающемуся тем, что он, кроме того, сверхпродуцирует :
- ген, который может быть получен за счет амплификации путем полимеразной цепной реакции при использовании пары праймеров, имеющих следующие последовательности:
5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID N° 138) и
5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID N° 139),
и в качестве матрицы космиды pSPM36 или полной ДНК Streptomyces ambofaciens, более предпочтительно, речь идет о гене, соответствующем кодирующей последовательности SEQ ID № 141,
- или ген, происходящий от них в соответствии с вырожденностью генетического кода.
Пример последовательности такого гена представлен в виде последовательности SEQ ID № 111 (ДНК), однако, эта последовательность является частичной, поскольку она не включает часть 3' соответствующей кодирующей последовательности. Трансляция протеина этой части кодирующей последовательности представлена в виде последовательности SEQ ID № 112. Специалист в данной области легко может ее дополнить, в частности, используя указание, представленное в примере 24. Затем была определена последовательность, не определенная в последовательности SEQ ID № 111, и полная последовательность этой открытой рамки считывания (orf28с) представлена в виде последовательности SEQ ID № 141. Трансляция протеина этой кодирующей последовательности представлена в виде последовательности SEQ ID № 142. В примере 24 представлен способ клонирования гена orf28c и получения экспрессирующего вектора, позволяющего осуществлять экспрессию orf28c. В этом примере также показано, что сверхпродуцирование гена orf28c в штамме OSC2 приводит к повышению продуцирования этим штаммом спирамицинов. Сверхпродуцирование гена orf28c может быть достигнуто путем увеличения числа копий этого гена и/или путем введения более активного промотора, чем исходный промотор. Предпочтительно, сверхпродуцировние вышеуказанного гена достигается за счет введения в микроорганизм конструкции рекомбинантной ДНК, позволяющей осуществлять сверхпродуцирование этого гена. Предпочтительно, эта конструкция рекомбинантной ДНК увеличивает число копий вышеуказанного гена и позволяет достигать сверхпродуцирования вышеуказанного гена. В этой конструкции рекомбинантной ДНК, последовательность, кодирующая ген, может находиться под контролем более активного промотора, чем исходный промотор. В качестве примера, можно назвать активный промотор ptrc, имеющийся в случае Streptomyces ambofaciens (Amann E. идр., 1988), а также промотор ermE*. Так, введенная копия или копии гена orf28c предпочтительно находятся под контролем промотора ermE*, как это имеет место в конструкции pSPM75 (см. пример 24).
Изобретение также относится к микроорганизму, продуцирующему спирамицин I, но не продуцирующему спиромицинов II и III, такому, как описанный выше, отличающемуся тем, что он сверхпродуцирует, кроме того, ген, соответствующий кодирующей последовательности SEQ ID № 47 или кодирующей последовательности, происходящей от нее в соответствии с вырожденностью генетического кода. Этот микрорганизм предпочтительно отличается тем, что речь идет о штамме SPM502 pSPM525, депонированном в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 26 февраля 2003 г. под регистрационным номером I-2977.
Изобретение также относится к способу продуцирования спиромицина I, причем способ состоит в культивировании, на соответствующей культуральной среде, микроорганизма, продуцирующего спирамицин I, но не продуцирующего спирамицины II и III, такого, как описанный выше, рекуперации кондиционированной культуральной среды или клеточного экстракта, выделении и очистке из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ного) на предыдущей стадии, спирамицина I. Условия культивирования такого микроорганизма могут быть определены согласно способам, хорошо известным специалистам в данной области. Культуральной средой может быть, например, среда МР5 или среда SL11 для Streptomyces и особенно для Streptomyces ambofaciens (Pernodet и др., 1993). Специалист, в частности, может обратиться к работе Kieser и др. (2000) в том, что касается культивирования Streptomyces. Продуцированный спирамицин I может быть рекуперирован любыми способами, известными спецалисту. Специалист может обратиться, например, к способам, указанным в патенте США 3000785, и, в особенности, к способам экстракции спирамицинов, описанным в этом патенте.
Другой объект изобретения относится к применению нуклеотидной последовательности согласно изобретению для повышения продуцирования макролидов микроорганизмом. Так, изобретение относится к мутантному, являющемуся продуцентом макролида микроорганизму, отличающемуся тем, что он имеет генетическую модификацию по меньшей мере в одном гене, включающем последовательность, такую, как определенная выше, и/или что она сверхпродуцирует по меньшей мере один ген, включающий последовательность, такую, как определенная выше. Генетическая модификация может состоять в супрессии, замене, делеции и/или инсерции одного или нескольких оснований в случае рассматриваемого гена или рассматриваемых генов с целью экспрессирования протеина или протеинов, обладающих высшей активностью, или экспрессирования на более высоком уровне этого или этих протеинов. Сверхпродуцирование рассматриваемого гена может быть достигнуто за счет увеличения числа копий этого гена и/или введения более активного промотора, чем исходный промотор. В качестве примера можно назвать активный промотор ptrc в случае Streptomyces ambofaciens (Amann E. и др., 1988), а также промотор ermE* (Bibb и др., 1985; Bibb и др., 1994). Так, сверхпродуцирование рассматриваемого гена может быть достигнуто путем введения в рассматриваемый микроорганизм, являющийся продуцентом макролида, конструкции рекомбинантной ДНК согласно изобретению, позволяющей осуществляться сверхпродуцированию этого гена. В самом деле, некоторые стадии биосинтеза макролидов являются лимитирующими и, если экспрессируют один или несколько более активных протеинов или если более значительный уровень экспрессии исходного протеина или исходных протеинов, участвующих на этих лимитирующих стадиях, можно повышать продуцирование соответствующего макролида или соответствующих макролидов. Увеличение степени продуцирования тилозина, например, в случае Streptomyces fradiae было достигнуто путем дупликации гена, кодирующего лимитирующую метилтрансферазу, превращающую макроцин в тилозин (Baltz R., 1997). Достижение экспрессии более активного протеина может быть осуществлено, в частности, путем мутагенеза, причем на этот предмет специалист может обратиться, например, к работе F. Ausubel и др. (2002). Предпочтительно, этими мутантными микроорганизмами, улучшенными в отношении продуцирования ими макролидов, являются бактерии рода Streptomyces. Более предпочтительно, рассматриваемым макролидом является спирамицин и микроорганизмами, в которых осуществляются мутагенез или мутагенезы, являются штаммы S. ambofaciens. Более предпочтительно, генетическую модификацию осуществляют в одном или нескольких генах, включающих одну из последовательностей, соответствующую одной или нескольким последовательностям SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 107, 109, 111, 113, 115, 118, 120, 141, 143, 145, 147 и 149 или один из ее вариантов или одну из последовательностей, происходящих от них в соответствии с вырожденностью генетического кода. Предпочтительно, микроорганизмы сверхпродуцируют один или несколько генов, включающих одну из последовательностей, соответствующую одной или нескольким последовательностям SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82, 84, 107, 109, 111, 113, 115, 118, 120, 141, 143, 145, 147 и 149 или один из ее вариантов или одну из последовательностей, происходящих от них в соответствии с вырожденностью генетического кода. Предпочтительно, микроорганизм сверхпродуцирует ген, включающий последовательность, соответствующую последовательности SEQ ID № 111 или 141, или один из ее вариантов или или одну из последовательностей, происходящих от них в соответствии с вырожденностью генетического кода. Представленная в виде SEQ ID № 111 последовательность является частичной, однако, специалист может легко дополнить ее, в частности, путем использования указания, данного в примере 24. Неопределенная последовательность в SEQ ID № 111 была затем определена и полная последовательность этой открытой рамки считывания (orf28c) представлена в виде SEQ ID № 141. Трансляция протеина этой кодирующей последовательности представлена в виде SEQ ID № 142. Так, в примере 24 представлен способ клонирования гена orf28c и получения экспрессирующего вектора, позволяющего осуществлять экпрессию orf28c. В этом примере также показано, что сверхпродуцирование гена orf28c в штамме OSC2 приводит к повышению продуцирования спирамицинов в этом штамме. Сверхпродуцирование гена orf28c может быть достигнуто за счет увеличения числа копий этого гена и/или за счет введения более активного промотора, чем исходный промотор. Предпочтительно, сверхпродуцирование гена orf28c достигается за счет введения в микроорганизм конструкции рекомбинантной ДНК, позволяющей осуществлять сверхпродуцирование этого гена. Предпочтительно, эта конструкция рекомбинантной ДНК увеличивает число копий гена orf28c и позволяет достигать сверхпродуцирования гена orf28c. В этой конструкции рекомбинантной ДНК, последовательность, кодирующая ген orf28c, может находиться под контролем более активного промотора, чем исходный промотор. В качестве примера, можно назвать активный промотор ptrc, имеющийся в случае Streptomyces ambofaciens (Amann E. идр., 1988), а также промотор ermE*. Так, введенная копия или копии гена orf28c предпочтительно находятся под контролем промотора ermE*, как это имеет место в конструкции pSPM75 (см. пример 24).
Другой аспект изобретения относится к способу продуцирования макролидов при использовании микрорганизмов, описанных в предыдущем абзаце. Этот способ состоит в культивировании, на соответствующей культуральной среде, микроорганизма, определенного в предыдущем абзаце, рекуперации кондиционированной культуральной среды или клеточного экстракта, выделении и очистке из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ного) на предыдущей стадии, продуцированного макролида или продуцированных макролидов. Условия культивирования таких микроорганизмов могут быть определены согласно способам, хорошо известным специалистам в данной области. Культуральной средой, например, может быть среда МР5 или среда SL11 для Streptomyces и особенно для Streptomyces ambofaciens (Pernodet и др., 1993). Специалист может обратиться, в частности, к работе Kieser и др. (2000) в том, что касается культивирования Streptomyces. Продуцированный макролид или продуцированные макролиды могут быть рекуперированы любыми способами, известными специалистам в данной области. Специалист может обратиться, например, к способам, указанным в патенте США 3000785, и в особенности к способам экстракции спирамицинов, описанным в этом патенте. Предпочтительно, микроорганизмами, используемыми в таком способе, являются бактерии рода Streptomyces. Более предпочтительно, рассматриваемым макролидом является спирамицин и мутантными микроорганизмами, улучшенными в отношении продуцирования ими спирмицинов, являются штаммы S. ambofaciens.
Следующий аспект изобретения относится к применению последовательности и/или вектора согласно изобретению для получения гибридных антибиотиков. В самом деле, полинуклеотиды согласно изобретению могут служить для получения микроорганизмов, экспрессирующих один или несколько мутантных протеинов, дающих возможность осуществлять модификацию в специфичности субстрата или еще которые могут быть экспрессированы в многочисленных микроорганизмах, являющихся продуцентами антибиотиков, с целью генерирования гибридных антибиотиков. Так, полинуклеотиды согласно изобретению, путем переноса генов между микроорганизмами-продуцентами, могут позволять получать гибридные антибиотики, обладающие интересными с фармакологической точки зрения свойствами (Hopwood и др., 1985а; Hopwood и др., 1985b; Hutchinson и др., 1989). Принцип, по которому генная инженерия может приводить к продуцированию гибридных антибиотиков, сначала был предложен Hopwood (Hopwood, 1981). Так, он предположил, что ферменты, участвующие в биосинтезе антибиотиков, часто акцептуют структурно связанные субстраты, но отличные от их нативного субстрата. Как правило, допускают (Hopwood,1981; Hutchinson, 1988; Robinson, 1988), что ферменты, кодируемые генами пути биосинтеза антибиотиков, обладают менее строгой специфичностью к субстрату, чем ферменты первичного метаболизма. Так, показано, что большое число ненативных субстратов трансформируется продуцирующими антибиотик микроорганизмами, их мутантами или очищенными ферментами пути биосинтеза этих антибиотиков (Hutchinson, 1988). Используя это указание, специалист в данной области может сконструировать микроорганизмы, экспрессионные один или несколько мутантных протеинов, позволяющих осуществлять модификацию в специфичности субстрата, с целью генерирования гибридных атибиотиков.
Изобретение также относится к применению по меньшей мере одного полинуклеотида и/или по меньшей мере одной рекомбинантной ДНК, и/или по меньшей мере одного экспрессирующего вектора, и/или по меньшей мере одного полипептида, и/или по меньшей мере одной клетки-хозяина, согласно изобретению, для осуществления одной или нескольких биоконверсий. Так, изобретение позволяет конструировать бактериальные или грибковые штаммы, в которых под контролем соответствующих сигналов экспрессии экспрессируются один или несколько протеинов согласно изобретению. Такие штаммы тогда могут быть использованы для осуществления биоконверсии или биоконверсий. Эти биоконверсии могут осуществляться либо с помощью целых клеток, либо с помощью клеточных экстрактов из указанных клеток. Эти биоконверсии могут позволять осуществлять трансформацию молекулы в производную форму с помощью фермента пути биосинтеза. Например, Carreras и др. описывают применение штамма Saccharopolyspora erythraea и штамма Streptomyces coelicolor для продуцирования новых производных эритромицинов (Carreras и др., 2002). Walczar и др. описывают применение монооксигеназы Р450 Streptomyces для биоконверсии дезацетиладриамицина (аналога антрациклина) в новые антрациклины (Walzcar и др., 2001). Olonao и др. описывают применение модифицированного штамма Streptomyces lividans для биоконверсии родомицинон-эпсилона в родомицин D (Olonao и др.,1999). Специалист может применять этот принцип в случае любого медиатора биосинтеза.
Изобретение относится также к рекомбинантной ДНК, отличающейся тем, что она включает:
- полинуклеотид, который может быть получен за счет амплификации путем полимеразной цепной реакции при использовании пары праймеров, имеющих следующие последовательности: 5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID № 138) и 5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID № 139), и в качестве матрицы космиды pSPM36 или полной ДНК Streptomyces ambofaciens, более предпочтительно речь идет о полинуклеотиде, соответствующем последовательности SEQ ID № 141;
- или фрагмент по меньшей мере из 10, 12, 15, 18, 20-25, 30, 40, 50, 60, 70, 80, 90, 100, 150, 200, 250, 300, 350, 400, 450, 500, 550, 600, 650, 700, 750, 800, 850, 900, 950, 1000, 1050, 1100, 1150, 1200, 1250, 1300, 1350, 1400, 1450, 1460, 1470, 1480, 1490 или 1500 последовательных нуклеотидов этих полинуклеотидов.
Предпочтительно, эта рекомбинантная ДНК представляет собой вектор. Еще более предпочтительно, вектор выбирают из бактериофагов, плазмид, фагемид, интегрирующих векторов, фосмид, космид, шаттл-векторов, ВАС (бактериальная искусствнная хромосома) или РАС (Р1-производная искусственная хромосома). В качестве пояснения, можно назвать в качестве бактериофагов фаг лямбда и фаг М13. В качестве плазмид можно назвать плазмиды, реплицирующиеся в E.coli, например, pBR322 и ее производные, pUC18 и ее производные, pUC19 и ее производные, рGB2 и ее производные (Churchward G. и др., 1984), pACYC177 (номер доступа в банк генов: Х06402) и ее производные, pACYC184 (номер доступа в банк генов: Х06403) и ее производные. Можно также назвать плазмиды, реплицирующиеся в Streptomyces, как, например, pIJ101 и ее производные, pSG5 и ее производные, SLP1 и ее производные, SCP2* и ее производные (Kieser и др., 2000). В качестве фагемид можно назвать, в качестве пояснения, pBluescript II и ее производные (выпускаются, в частности, фирмой Stratagene (LaJolla, Калифорния, США)), pGEM-T и ее производные (выпускается фирмой Promega (Мэдисон, Висконсин, США)), λZAPII и ее производные (выпускаются, в частности, фирмой Stratagene (LaJolla, Калифорния, США)). В качестве интегрирующих векторов можно назвать, в качестве пояснения, интегрирующие векторы в случае Streptomyces, как, например, таковые, происходящие от SLP1 (Kieser и др., 2000), таковые, происходящие от pSAM2 (Kieser и др., 2000), векторы, использующие системы интеграции фагов PhiC31 (Kieser и др., 2000) (например, pSET152 (Bierman и др., 1992)) или VWB (Van Mellaert L. и др., 1998), а также векторы, использующие систему интеграции IS117 (Kieser и др., 2000). В качестве фосмид можно назвать, в качестве пояснения, фосмиду pFOS1 (выпускается фирмой New England Biolabs Inc., Beverly, Massachussetts, США) и ее производные. В качестве космид можно назвать, в качестве пояснения, космиду SuperCos и ее производные (выпускаются, в частности, фирмой Stratagene (LaJolla, Калифорния, США)), космиду pWED15 (Wahl и др., 1987) и ее производные. В качестве шаттл-векторов можно назвать, в качестве пояснения, шаттл-плазмиды E.coli/Streptomyces, как, например, pIJ903 и ее производные, ряд пдазмид pUWL, pCAO106, pWHM3, pOJ446 и их производные (Kieser и др., 2000), челночные ВАС E.coli/Streptomyces, как, например, таковые, описанные в Международной заявке на патент WO-01/40497. В качестве ВАС (бактериальная искусственная хромосома) можно назвать, в качестве пояснения, ВАС pBeloBAC11 (номер доступа в банк генов: U51113). В качестве РАС (Р1-производная искусственная хромосома) можно назвать, в качестве пояснения, вектор pCYPAC6 (номер доступа в банк генов: AF133437). Более предпочтительно, этой рекомбинантной ДНК является экспрессирующий вектор. Экспрессионные векторы, используемые в различных экспрессирующих системах, хорошо известны специалистам в данной области; в том, что касается прокариотных клеток, можно назвать, в качестве пояснения, экспрессионные векторы в случае в E.coli, например, семейства рЕТ, выпускаемые фирмой Stratagene (LaJolla, Калифорния, США), векторы семейства GATEWAY, выпускаемые фирмой Invitrogen (Carlsbad, Калифорния, США), векторы семейства pBAD, выпускаемые фирмой Invitrogen (Carlsbad, Калифорния, США), векторы семейства pMAL, выпускаемые фирмой New England Biolabs Inc. (Beverly, Massachussetts, США), экспрессионные векторы, индуктируемые рамнозой, указанные в публикации Wilms B. и др., 2001, и их производные, можно также назвать экспрессионные векторы в случае Streptomyces, как, например, векторы pIJ4123, pIJ6021, pPM927, pANT849, pANT850, pANT851, pANT1200, pANT1201, pANT1202, и их производные (Kaiser и др., 2000). В том, что касается дрожжевых клеток, можно назвать, в качестве пояснения, вектор pESC, выпускаемый фирмой Stratagene (LaJolla, Калифорния, США). В том, что касается бакуловирусной экспрессионной системы, позволяющей осуществлять экспрессию в клетках насекомых, можно назвать, в качестве пояснения, вектор ВасРАК6, выпускаемый фирмой BD Biosciences Clontech (Palo Alto, Калифорния, США). В том, что касается клеток млекопитающих, можно назвать, в качестве примера, векторы, включающие промотор ранних генов вируса CMV (цитомегаловирус) (например, вектор pCMV и его производные выпускаются фирмой Stratagene (LaJolla, Калифорния, США)), или промотор предранних генов (ранний промотор SV40) вакуолизирующего обезьяньего вируса SV40 (например, вектор pSG5, выпускаемый фирмой Stratagene (LaJolla, Калифорния, США). Следующий аспект изобретения относится к клеткам-хозяевам, в которые введена по меньшей одна рекомбинантная ДНК, описанная в данном абзаце.
Следующий аспект изобретения относится к способу продуцирования полипептида, отличающемуся тем, что вышеуказанный способ включает следующие стадии:
а) трансформация клетки-хозяина с помощью по меньшей мере одного экспрессирующего вектора, такого, как описанный в вышеприведенном абзаце;
b) культивирование, на соответствующей культуральной среде, вышеуказанной клетки-хозяина;
с) рекуперация кондиционированной культуральной среды или клеточного экстракта;
d) выделение и очистка из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ного) на стадии с), вышеуказанного полипептида;
е) в желательном случае, охарактеризовывание полученного рекомбинантного полипептида.
Продуцированный таким образом рекомбинантный полипептид может быть очищен путем пропускания через соответствующий ряд хроматографических колонок согласно способам, известным специалистам в данной области и описанным, например, F. Ausubel и др. (2002). В качестве примера можно назвать способ с гистидиновой меткой (“Tag-Histidine”), который состоит в добавлении короткой полигистидиновой последовательности к продуцируемому полипептиду, причем этот последний затем может быть очищен на никелевой колонке. Этот полипептид также может быть получен способами синтеза in vitro. В качестве пояснения таких способов, полипептид может быть получен с помощью системы, называемой как “система быстрой трансляции (RTS)”, выпускаемой, в частности, фирмой Roche Diagnostics France S.A., Meylan, Франция.
Другой аспект изобретения относится к микроорганизму, продуцирующему по меньшей мере один спирамицин, отличающемуся тем, что он сверхпродуцирует :
- ген, который может быть получен за счет амплификации путем полимеразной цепной реакции (PCR) при использовании пары праймеров, имеющих следующие последовательности: 5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID № 138) и 5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID № 139), и в качестве матрицы космиды pSPM36 или полной ДНК Streptomyces ambofaciens, более предпочтительно, речь идет о гене, соответствующем кодирующей последовательности SEQ ID № 141,
- или ген, происходящий от него в соответствии с вырожденностью генетического кода.
Пример последовательности такого гена представлен в виде последовательности SEQ ID № 111 (ДНК), однако, эта последовательность является частичной, поскольку она не включает часть 3' соответствующей кодирующей последовательности. Трансляция протеина этой части кодирующей последовательности представлена в виде последовательности SEQ ID № 112. Специалист в данной области легко может ее дополнить, используя, в частности, указание, представленное в примере 24. В этом примере также представлен способ клонирования гена orf28c и получения экспрессирующего вектора, позволяющего осуществлять экспрессию orf28c. В этом примере также показано, что сверхпродуцирование гена orf28c в штамме OSC2 приводит к повышению продуцирования этим штаммом спирамицинов. Предпочтительно, микроорганизмом, сверхпродуцирующим
- ген, который может быть получен за счет амплификации путем полимеразной цепной реакции (PCR) при использовании пары праймеров, имеющих следующие последовательности: 5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID № 138) и 5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID № 139), и в качестве матрицы космиды pSPM36 или полной ДНК Streptomyces ambofaciens, более предпочтительно, речь идет о гене, соответствующем кодирующей последовательности SEQ ID № 141,
- или ген, происходящий от него в соответствии с вырожденностью генетического кода,
является бактерия рода Streptomyces, еще более предпочтительно, речь идет о бактерии вида Streptomyces ambofaciens. Предпочтительно, сверхпродуцирование вышеуказанного гена достигается путем трансформации вышеуказанного микроорганизма с помощью экспрессирующего вектора, в высшей степени предпочтительно, штаммом микроорганизма является штамм OSC2/pSPM75(1) или штамм OSC2/pSPM(2), депонированный в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue Docteur Roux, 75724 Париж, Cedex 15, Франция, 6 октября 2003 г. под регистрационным номером I-3101.
Следующий аспект изобретения относится к способу продуцирования спиромицина (спирамицинов) при использовании микроорганизмов, описанных в предыдущем абзаце. Этот способ состоит в культивировании, на соответствующей культуральной среде, микроорганизма, определенного в предыдущем абзаце, рекуперации кондиционированной культуральной среды или клеточного экстракта, выделении и очистке из вышеуказанной культуральной среды или еще из клеточного экстракта, полученной(ного) на предыдущей стадии, продуцированного спирамицина или продуцированных спирамицинов. Условия культивирования таких микроорганизмов могут быть определены согласно способам, хорошо известным специалистам в данной области. Культуральной средой может быть, например, среда МР5 или среда SL11 для Streptomyces и особенно для Streptomyces ambofaciens (Pernodet и др., 1993). Специалист, в частности, может обратиться к работе Kieser и др. (2000) в том, что касается культивирования Streptomyces. Продуцированный спирамицин или продуцированные спирамицины могут быть рекуперированы любыми способами, известными спецалистам в данной области. Специалист может обратиться, например, к способам, указанным в патенте США 3000785, и, в особенности, к способам экстракции спирамицинов, описанным в этом патенте.
Предпочтительно, микроорганизмами, используемыми в таком способе, являются бактерии рода Streptomyces. Более предпочтительно, микроорганизмами являются штаммы S.amboficiens.
Другой аспект изобретения относится к экспрессирующему вектору, отличающемуся тем, что полинуклеотид, соответствующий последовательности SEQ ID № 47, или полинуклеотид, происходящий от него в соответствии с вырожденностью генетического кода, находится под контролем промотора, позволяющего осуществлять экспрессию протеина, кодируемого упомянутым нуклеотидом в случае Streptomyces ambofaciens. Примеры экспрессирующих векторов, используемых в случае Streptomyces, даны выше. Такой экспрессирующий вектор предпочтительно отличается тем, что речь идет о плазмиде pSPM524 или pSPM525.
Следующий аспект изобретения относится к штамму Streptomyces ambofaciens, трансформированному с помощью вектора, определенного в предыдущем абзаце.
Другой аспект изобретения относится к полипептиду, отличающемуся тем, что его последовательность включает последовательность SEQ ID № 112. Изобретение также относится к полипептиду, отличающемуся тем, что его последовательность соответствует последовательности, выражаемой кодирующей последовательностью:
- гена, который может быть получен за счет амплификации путем полимеразной цепной реакции (PCR) при использовании пары праймеров, имеющих следующие последовательности: 5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID № 138) и 5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID № 139), и в качестве матрицы космиды pSPM36 или полной ДНК Streptomyces ambofaciens, более предпочтительно, речь идет о гене, соответствующем кодирующей последовательности SEQ ID № 141,
- или гена, происходящего от него в соответствии с вырожденностью генетического кода.
Предпочтительно, эти полипептиды экспрессируются в нативном состоянии бактерией рода Streptomyces, более предпочтительно, эти полипептиды участвуют в биосинтезе спирамицинов.
Другой аспект изобретения относится к экспрессирующему вектору, позволяющему осуществлять экспрессию полипептида, такого, как определенный в предыдущем абзаце, в Streptomyces ambofaciens. Примеры используемых экспрессирующих векторов в случае Streptomyces даны выше. Рассматриваемым экспрессирующим вектором предпочтительно является плазмида pSPM75.
Список фигур
Фиг.1: Химическая структура спирамицинов I, II и III.
Фиг.2: Космиды, используемые для секвенирования области.
Фиг.3: Организация группы генов, участвующих в пути биосинтеза спирамицинов.
Фиг.4: Предполагаемый путь биосинтеза микарозы.
Фиг.5: Предполагаемый путь биосинтеза микаминозы.
Фиг.6: Предполагаемый путь биосинтеза форозамина.
Фиг.7: Предпочтительный порядок введения сахаров в молекулу спирамицина и медиаторы.
Фиг.8: Предполагаемый путь биосинтеза метоксималонила в случае S.ambofaciens. Этот путь предполагается по аналогии с биосинтезом метоксималонила в случае Streptomyces hygroscopicus var. ascomyceticus (Wu K. и др., 2000).
Фиг.9: Стадии, приводящие к инактивации гена:
А) клонирование гена-мишени в векторе, реплицирующемся в E.coli, но не в Streptomyces;
В) инсерция кластера резистентности в ген-мишень (путем клонирования или рекомбинации между идентичными короткими последовательностями);
С) введение плазмиды в Streptomyces ambofaciens (путем трансформации или конъюгации с E.coli) и селекция клонов с интегрированным кластером, затем просеивание клонов, утративших часть вектора, для получения возможности замены гена;
D) область хромосомы мутантного штамма, в котором ген-мишень инактивирован путем замены гена.
Фиг.10: Необязательные стадии, следующие за инактивацией гена по способу, описанному согласно фиг.9, которые могут быть осуществлены, если для прерывания гена-мишени был использован эксцизируемый кластер:
Е) введение в мутантный штамм плазмиды pOSV508, несущей гены xis и int pSAM2, вырабатываемые которыми продукты позволяют осуществлять эффективную эксцизию путем специфической рекомбинации сайтов между последовательностями attL и attR, фланкирующими кластер;
F) получение клонов, утративших эксцизируемый кластер и чувствительных к антибиотику, к которому кластер придал резистентность;
G) после роста и споруляции на твердой среде без антибиотика, с высокой частотой утрачивается плазмида pOSV508. Таким образом можно получать клоны, чувствительные к тиострептону, в которых ген-мишень содержить делецию в открытой рамке считывания. Последовательность подвергнутого делеции гена-мишени может контролироваться за счет амплификации путем РСR и секвенирования продукта PCR.
Фиг.11: Амплификация эксцизируемого кластера с целью использования для эксперимента по гомологичной рекомбинации. Технология гомологичной рекомбинации за счет коротких гомологичных последовательностей описана Chaveroche и др. (2000). 39 или 40 Дезоксинуклеотидов, расположенных у конца 5' этих олигонуклеотидов, включают последовательность, соответствующую последовательности инактивируемого гена, и 20 дезоксинуклеотидов, расположенных ближе к концу 3', соответствуют последовательности одного из концов эксцизируемого кластера.
Фиг.12: Получение конструкции для инактивации гена-мишени по способу, описанному Chaveroche и др. (2000).
Фиг.13: Карта плазмиды pWHM3. Strepto ori: начало репликации Streptomyces.
Фиг.14: Карта плазмиды pOSV508.
Фиг.15: Пример структуры эксцизируемого кластера. Он образован кластером Ωhyg (Blondelet-Rouault и др., 1997), фланкированным сайтами attR и attL (Raynal и др., 1998), рекомбинация между которыми позволяет осуществлять эксцизию кластера за счет экспрессии генов xis и int.
Фиг.16: Карта плазмиды pBXL1111.
Фиг.17: Карта плазмиды pBXL1112.
Фиг.18: Микробиологический тест на продуцирование спирамицина, основанный на чувствительности штамма Micrococcus luteus к спирамицину. Используемый штамм Micrococcus luteus представляет собой штамм, по своей природе чувствительный к спирамицину, но резистентный к конгоцидину. Различные тестируемые штаммы Streptomyces культивировали в колбах Эрленмейера емкостью 500 мл, содержащих 70 мл среды MP5, при инокуляции с помощью начальной концентрации 2,5·106 спор/мл и выращивании при температуре 27°С при орбитальном перемешивании со скоростью 250 об/мин. Отборы проб ферментационного сусла осуществляли после 48, 72 и 96 часов культивирования и цетрифугировали. Для теста использовали разведение в десять раз этих супернатантов в стерильной культуральной среде. Индикаторный штамм Micrococcus luteus, резистентный к конгоцидину, но чувствительный к спирамицину (Gourmelen и др., 1998), культивировали в квадратной чашке размером 12 см х 12 см. Диски из бумаги Whatman AA пропитывали с помощью 70 мкл десятикратного разведения каждого супернатанта и помещали на поверхность чашки. Диски, пропитанные раствором спирамицина различных концентраций (2-4-8 мкг/мл культуральной среды МР5), использовали в качестве эталонного ряда. Чашки инкубировали при температуре 37°С в течение 24-48 часов. Если диск содержит спирамицин, то он диффундирует в агар и ингибируют рост индикаторного штамма Micrococcus luteus. Это ингибирование вызывает образование “гало” вокруг диска, причем это гало отражает зону, где штамм Micrococcus luteus не растет. Наличие этого гало, следовательно, является признаком наличия спирамицина и позволяет определять, продуцирует ли или не продуцирует спирамицин рассматриваемый штамм S.ambofaciens. Сравнение с диаметрами ингибирования, достигаемыми в случае эталонного ряда, позволяет получить указание на количество спирамицина, продуцированного этим штаммом.
Фиг.19: Хроматограмма ВЭЖХ супернатанта, отфильтрованного от культуральной среды штамма OSC2.
Фиг.20: Хроматограмма ВЭЖХ супернатанта, отфильтрованного от культуральной среды штамма SPM501.
Фиг.21: Хроматограмма ВЭЖХ супернатанта, отфильтрованного от культуральной среды штамма SPM502.
Фиг.22: Хроматограмма ВЭЖХ супернатанта, отфильтрованного от культуральной среды штамма SPM507.
Фиг.23: Хроматограмма ВЭЖХ супернатанта, отфильтрованного от культуральной среды штамма SPM508.
Фиг.24: Хроматограмма ВЭЖХ супернатанта, отфильтрованного от культуральной среды штамма SPM509.
Фиг.25: Сопоставление путем выравнивания протеина Orf3 (SEQ ID № 29) c протеином TylB (SEQ ID № 87) S. fradiae, осуществляемое согласно программе FASTA.
Фиг.26: Сопоставление путем выравнивания протеина MdmA S. mycarofaciens (SEQ ID № 88) c протеином SrmD (SEQ ID № 16), осуществляемое согласно программе FASTA.
Фиг.27: Пример последовательностей остаточных сайтов после эксцизии эксцизируемого кластера. Жирным шрифтом указан минимальный сайт att26, такой, как определенный Raynal и др., 1998. Последовательность открытой рамки считывания 1 (att1) из 33 нуклеотидов представлена в виде последовательности SEQ ID № 104, последовательность открытой рамки считывания 2 (att2) из 34 нуклеотидов представлена в виде последовательности SEQ ID № 105, последовательность открытой рамки считывания 3 (att3) из 35 нуклеотидов представлена в виде последовательности SEQ ID № 95.
Фиг.28: Схематическое изображение гена orf10, локализация используемых праймеров PCR и конструкция, получаемая с каждой парой праймеров.
Фиг.29: Конструкция кластера рас-oritT.
Фиг.30: Карта космиды pWED2.
Фиг.31: Схематическое изображение группы генов, участвующих в пути биосинтеза спирамицинов и локализация трех зондов, используемых для выделения космид из банка геномных ДНК штамма OSC2 Streptomyces ambofaciens в случае E.coli (см. пример 19).
Фиг.32: Локализация вставок космид, выделенных из банка геномных ДНК штамма OSC2 Streptomyces ambofaciens в случае E.coli (см. пример 19). Новый банк космидных ДНК.
Фиг.33: Субклонирование фрагмента PstI-PstI космиды pSPM36 (вставка плазмиды pSPM58), субклонирование фрагмента StuI-StuI космиды pSPM36 (вставка плазмиды pSPM72), субклонирование фрагмента EcoRI-StuI (вставка плазмиды pSPM73).
Фиг.34: Локализация открытых рамок считывания, идентифицированных во вставке PstI-PstI плазмиды pSPM58 и во вставке EcoRI-StuI плазмиды pSPM73.
Фиг.35: Суперпозиция хроматограмм ВЭЖХ супернатанта, отфильтрованного от культуральной среды штамма OS49.67, полученных при 238 нм и 280 нм (вверху), и УФ-спектров молекул, элюированных в случае 33,4 минут и 44,8 минут (внизу).
Фиг.36: Молекулярная структура молекул платенолида А и платенолида В.
Фиг.37: Организация группы генов, участвующих в пути биосинтеза спирамицинов.
Фиг.38: Молекулярная структура медиатора биосинтеза, продуцируемого штаммом SPM507.
Фиг.39: Структура медиатора биосинтеза, продуцируемого штаммом S. ambofaciens генотипа orf6*::att1Ωhyg+, получаемого из штамма, сверхпродуцирующего спирамицины. Инсерция кластера att1Ωhyg+ в orf6* вызывает полярный эффект, предотвращающий экспрессию orf5*.
Фиг.40: Молекулярная структура молекул платенолид А + микароза и платенолид В + микароза, продуцируемых штаммом OS49.67.
Фиг.41: Субклонирование фрагмента PstI-PstI космиды pSPM36 (вставка плазмиды (pSPM79)), локализация открытых рамок считывания, идентифицированных во вставке PstI-PstI плазмиды рSPM79 и локализация последовательности SEQ ID № 140.
Настоящее изобретение поясняется с помощью нижеследующих примеров, которые нужно рассматривать как пояснительные, а не ограничивающие объем охраны изобретения.
Вкратце, гены пути биосинтеза спирамицинов выделяли из геномного банка ДНК Streptomyces ambofaciens. Этот банк получали путем частичной рестрикции геномной ДНК Streptomyces ambofaciens ферментом рестрикции BamHI. Большие фрагменты ДНК, в среднем длиной 35-45 т.н., клонировали в космиде pWED1 (Gourmelen и др., 1998), происходящей от космиды pWED15 (Wahl и др., 1987). Эти космиды вводили в E.coli благодаря фаговым частицам. Таким образом полученный банк подвергали гибридизации с зондом (соответствующим последовательности SEQ ID № 86), соответствующим части гена tylB S. fradiae (Merson-Davies & Cundliffe, 1994; номер доступа в банк генов: U08223). После гибридизации в особенности отбирали космиду по 4 гибридизирующимся с зондом областям. Эту космиду, названную pOS49.1, затем рестриктировали с помощью SacI и фрагмент длиной 3,3 т.н., содержащий область, гибридизирующуся с зондом, клонировали в векторе pUC19 и секвенировали. Были идентифицированы четыре открытые рамки считывания и одна из них кодирует протеин (SEQ ID № 29), обладающий большими сходствами последовательности с протеином TylB S. fradiae (SEQ ID № 87) (cм. Фиг.25). Этот ген был назван orf3 (SEQ ID № 28) и инактивирован в S. ambjfaciens. Показано, что инактивированные в гене orf3 клоны не продуцируют больше спирамицинов. Это свидетельствует об участии гена orf3 или расположенных ниже генов в биосинтезе спирамицинов.
На основании этого полученного подтверждения секвенировали гораздо больший участок космиды pOS49.1 с той и другой стороны исследованного ранее фрагмента SacI. Так, из космиды pOS49.1 получали последовательность области, включающей семь полных открытых рамок считывания и две другие неполные открытые рамки считывания, расположенные с той и другой стороны этих семи полных открытых рамок считывания. За счет поиска в базах данных показано, что одна из неполных открытых рамок считывания соответствует локусу srmG (область, кодирующая фермент, называемый как “поликетидсинтаза” (PKS)). Соответствующие гены были клонированы согласно Burgett S. и др. в 1996 г. (патент США 5945320). Кроме того, в базах данных не были обнаружены другие открытые рамки считывания: семь полных открытых рамок считывания, названных orf1, orf2, orf3, orf4, orf5, orf6, orf7 (SEQ ID № 23, 25, 28, 30, 34, 36, 40), и начало восьмой открытой рамки считывания, названной orf8 (последовательность SEQ ID № 43).
Во-вторых, и с целью клонирования других генов, участвующих в биосинтезе спирамицинов, в той же самой области были выделены другие космиды, включающие фрагменты генома S. ambofaciens. Для этого осуществляли новую серию гибридизаций в колониях, используя три зонда. Первый зонд соответствует фрагменту ДНК длиной 3,7 т.н., содержащему orf1, orf2 и начало orf3, субклонированному из pOS49.1. Второй зонд соответствует фрагменту длиной 2 т.н. ДНК, содержащему часть prf7 и часть orf8 и субклонированному из pOS49.1. Также был использован третий зонд. Этот последний соответствует фрагменту ДНК длиной 1,8 т.н., содержащму ген srmD. Ген srmD представляет собой ген, выделенный из S. ambofaciens, способный придавать резистентность к спирамицину. В самом деле, согласно предыдущим работам, осуществили клонирование нескольких детерминант резистентности S. ambofaciens, придающих резистентность к спирамицину, в штамме S. griseofuscus (штамм, чувствительный к спирамицину) (Pernodet и др., 1993; Pernodet и др., 1999). Для выделения генов резистентности был реализован космидный банк геномных ДНК штамма S. ambofaciens в космиде pKC505 (Richardson M.A. и др., 1987). Этот пул космид был введен в S. griseofuscus, по своей природе чувствительный к спирамицину. Таким образом были получены пять космид, способных придавать резистентность к апрамицину и спирамицину штамму S. griseofuscus. Среди этих 5 космид, одна космида, названная pOS44.1, имеет в своей вставке ген srmD, который кодирует протеин, обладающий некоторым сходством с протеином, кодируемым геном mdmA Streptomyces mycarofaciens и участвующим в резистентности к мидекамицину в этом организме-продуценте (Hara и др., 1990; номер доступа в банк генов; А60725) (фиг.26). Третий зонд, используемый для локализации генов биосинтеза спирамицина, представляет собой вставку длиной примерно 1,8 т.н., включающую ген srmD.
Эти три зонда были использованы для гибридизации банка геномных ДНК, описанного выше, и позволили выбрать две космиды (pSPM7 и pSPM5), способные содержать самые длинные вставки и не имеющие общих зон. Космида pSPM5 гибридизуется с первым и вторым зондом, но не гибридизуется с третьим зондом, тогда как космида pSPM7 гибридизуется только с третьим зондом. Эти две космиды полностью секвенировали методом “выстрела из дробового ружья”. Последовательности вставок этих двух космид pSPM7 и pSPM5 могут быть соединены, так как, хотя и не перекрываются, каждая из вставок включает известную на одном из ее концов последовательность. В самом деле, каждая из этих вставок включает фрагмент последовательности одного из генов, кодирующих фермент, называемый “поликетидсинтаза” (PKS). Эти 5 генов были клонированы Burgett S. и др. в 1996 г. (патент США 5945320) (см. фиг.2). Таким образом, была определена одна последовательность геномной ДНК S. ambafaciens. Последовательность из 30943 нуклеотидов, начинающаяся, в 5', с сайта EcoRI, расположенного в первом гене PKS, и доходящая вплоть до сайта BamHI в 3', представлена в виде последовательности SEQ ID № 1. Эта последовательность соответствует области выше генов PKS (см. фиг.2 и 3). Вторая область из 11171 нуклеотидов, начинающаяся с сайта PstI в 5' и доходящая вплоть до сайта BstEII в 3', расположенного в пятом гене PKS, представлена в виде последовательности SEQ ID № 2. Этой областью является область ниже генов PKS (причем низ и верх определяются ориентацией 5 генов PKS, которые все имеют одну и ту же смысловую ориентацию) (см. фиг.2 и 3).
В третьих, и с целью клонирования других генов, участвующих в биосинтезе спирамицинов, были выделены другие космиды, включающие фрагменты генома S. ambofaciens, в той же самой области.
Пример 1
Конструирование банка геномных ДНК штамма АТСС23877 Streptomyces ambofaciens в E.coli
1.1. Экстракция геномной ДНК из штамма АТСС23877 Streptomyces ambofaciens
Штамм АТСС23877 Streptomyces ambofaciens (доступный, в частности, из Американской коллекции типовых культур (АТСС) (Manassas, Virginie, США) под номером 23877)) культивировали на среде YEME (дрожжевой экстракт-солодовый экстракт) (Kieser T. и др., 2000) и геномную ДНК этого штамма экстрагировали и очищали согласно стандартным способам лизиса и преципитации (Kieser T. и др., 2000).
1.2. Конструирование банка геномных ДНК
Геномную ДНК штамма АТСС23877 Streptomyces ambofaciens, такую, как выделенная выше, частично рестриктировали ферментом рестрикции BamHI для получения фрагментов ДНК длиной от примерно 35 т.н. до 45 т.н. Эти фрагменты клонировали в космиде pWEDI (Gourmelen и др., 1998), предварительно рестриктированной с помощью BamHI. Космида pWED1 образуется от космиды рWED15 (Wahl и др., 1987) за счет делеции фрагмента HpaI-HpaI длиной 4,1 т.н., содержащего активный модуль экспрессии у млекопитающих (Gouemelan и др., 1998). Полученную в результате лигирования смесь затем инкапсидировали in vitro в частицы лямбда-фагов с помощью системы “Packagene® Lambda DNA packaging system”, выпускаемой фирмой Promega, согласно рекомендациям изготовителя. Полученные фаговые частицы использовали для инфицирования штамма SURE® E.coli, выпускаемого фирмой Stratagene (LaJolla, Калифорния, США). Селекцию клонов осуществляли на среде LB + ампициллин (50 мкг/мл), поскольку космиде pWED1 придают резистентность к ампициллину.
Пример 2
Выделение и характеристика генов, участвующих в биосинтезе спирамицинов в Streptomyces ambofaciens
2.1. Гибридизация в колонии клонов E.coli геномного банка Streptomyces ambofaciens АТСС23877
Примерно 2000 клонов E.coli полученного выше банка переносили на фильтр для гибридизации колоний. Используемым для гибридизации зондом является фрагмент NaeI-NaeI ДНК (SEQ ID № 86), включающий часть гена tylB Streptomyces fraidae. Этот фрагмент соответствует нуклеотидам 2663-3702 фрагмента ДНК, описанного Merson-Davies L.A. и Cundliffe E. (Merson-Davies L.A. & Cundliffe E., 1994; номер доступа в банк генов: U08223), в котором кодирующая последовательность гена tylB соответствует нуклеотидам 2677-3843.
Фрагмент NaeI-NaeI ДНК, несущий часть гена tylB Streptomyces fraidae (SEQ ID № 86), метили с помощью 32Р по способу “произвольного примирования” (набор, выпускаемый фирмой Roche) и использовали в качестве зонда для гибридизации 2000 клонов банка, перенесенных на фильтр. Используемыми мембранами являются нейлоновые мембраны Hybond N, выпускаемые фирмой Amersham (Amersham Biosciences, Orsay, Франция), и гибридизацию осуществляли при температуре 55°С в буфере, описанном Church и Gilbert (Church & Gilbert, 1984). Одну промывку осуществляли в 2Х SSC при температуре 55°С в течение 15 минут и две последовательные промывки осуществляли затем в 0,5Х SSC при температуре 55°С продолжительностью 15 минут каждая. В этих условиях гибридизации и промывки, 4 клона из 2000 гибридизированных обладали сильным сигналом гибридизации. Эти 4 клона культивировали на среде LB + ампициллин (50 мкг/мл) и соответствующие 4 кисмиды экстрагировали путем стандартного щелочного лизиса (Sambrook и др., 1989). Затем подтверждали, что гибридизация действительно имела место в фрагменте ДНК, присутствующем во вставке этих четырех космид. Для этого космиды рестриктировали независимо несколькими ферментами (BamHI, PstI и SacI). Продукты рестрикций разделяли на геле агарозы, переносили на мембрану Nylon и гибридизировали с помощью фрагмента NaeI-NaeI ДНК, включающего часть гена tylB Streptomyces fraidae (см. выше), в тех же условиях, что и указанные выше. Признаны действительными четыре космиды и предпочтительно была выбрана одна из этих космид и названа pOS49.1.
2.2. Подтверждение участия идентифицированной области и секвенирование вставки космиды pOS49.1
Субклонировали несколько фрагментов вставки космиды pOS49.1 и определяли их последовательности. Космиду pOS49.1 рестриктировали ферментом SacI и путем саузерн-блоттинга в вышеописанных условиях показано, что фрагмент длиной 3,3 т.н. содержит область, гибридизирующуюся с зондом tylB. Этот фрагмент длиной 3,3 т.н. выделяли путем электроэлюирования из 0,8%-ного геля агарозы, затем клонировали в векторе pUC19 (номер доступа в банк генов: M77789) и секвенировали. Таким образом полученная пазмида была названа pOS49.11. Четыре открытые рамки считывания с типичным использованием кодонов Streptomyces были идентифицированы в этом фрагменте (две полные и две укороченные) с помощью программы FramePlot (Ishikawa J. & Hotta K., 1999). Сравнения последовательностей по программе FASTA (см. Pearson W.R. & Lipman D.J., 1988; и Ptarson W.R., 1990), доступной, в частности, из центра ресурсов INFOBIOGEN, Evry, Франция), позволили обнаружить, что протеин, следующий из одной из этих 4 открытых рамок считывания, обладает большим сходством последовательности с протеином TylB (SEQ ID № 87; номер доступа в банк генов: U08223) S. fradiae (см. фиг.25). Этот протеин был назван Prf3 (SEQ ID № 29).
С целью тестирования, участвует ли соответствующий ген (ген orf3 (SEQ ID № 28)) в биосинтезе спирамицинов в случае S. ambofaciens, этот ген прерывали кластером Ωhyg (Blondelet-Rouault M.-H. и др., 1997; номер доступа в банк генов: Х99315). Для этого плазмиду pOS49.11 рестриктировали ферментом XhoI и фрагмент, содержащий четыре открытые рамки считывания (две полные и две укороченные, в которых orf3 находится целиком), субклонировали на уровне сайта XhoI вектора рВСSK +, выпускаемого фирмой Stratagene (LaJolla, Калифорния, США). Таким образом полученная плазмида была названа pOS49.12. С целью инактивации orf3, внутренний фрагмент PmlI-BstEII orf3 заменяли кластером Ωhyg путем клонирования свободного конца в этой последней плазмиде. Для этого плазмиду pOS49.12 рестриктировали ферментами PmlI и BstEII, единственный сайт которых находится в кодирующей последовательности гена orf3. Концы фрагмента, соответствующего вектору, делали свободными концами путем обработки ферментом Кленова (большой фрагмент ДНК-полимеразы I). Кластер Ωhyg получали путем рестрикции ферментом BamHI плазмиды pHP45 Ωhyg (Blondelet-Rouault и др., 1997; номер доступа в банк генов: Х99315). Фрагмент, соответствующий кластеру Ωhyg, рекуперировали на геле агарозы и его концы делали свободными концами путем обработки ферментом Кленова. Таким образом полученные свободные концы двух фрагментов (кластер Ωhyg и плазмида pOS49.12) лигировали и продукт лигирования использовали для трансформации бактерий E.coli. Таким образом полученная плазмида была названа pOS40.14 и она содержит ген orf3, прерванный кластером Ωhyg.
Вставку плазмиды pOS49.14, в виде фрагмента XhoI-XhoI, концы которого были сделаны нелипкими путем обработки ферментом Кленова, клонировали на уровне сайта EcoRV плазмиды pOJ260 (плазмида pOJ260 представляет собой конъюгативную плазмиду, способную реплицироваться в E.coli, но неспособную реплицироваться в S. ambofaciens (Bierman M. и др., 1992). Эта плазмида придает резистентность к апрамицину штаммам E.coli и Streptomyces). Полученная плазмида (вставка плазмиды pOS49.14, клонированная в плазмиде pOJ260) была названа pOS49.16. Эту последнюю переносили в штамм АТСС23877 S. ambofaciens за счет конъюгации с помощью конъюгативного штамма E.coli S17-1, как описанный Mazodier и др. (Mazodier и др., 1989). Штамм E.coli S17-1 происходит от штамма E.coli 294 (Simon и др., 1983; Simon и др., 1986). Получали трансконъюгирующиеся клоны, обладающие маркером резистентности к гигромицину, находящемуся в кластере Ωhyg, и утратившие маркер резистентности к апрамицину, находящийся в векторе pOJ260. Для этого клоны селекционировали по их резистентности к гигромицину. Резистентные к гигромицину клоны затем пересевали, соответственно, на среду с гигромицином (антибиотик В) и на среду с апрамицином (антибиотик А) (см. фиг.9). Клоны, резистентные к гигромицину (HygR) и чувствительные к апрамицину (ApraS), представляют собой, в принципе, такие, где произошло двойное явление рекомбинации и которые, следовательно, обладают геном orf3, прерванным кластером Ωhyg. Замену исходной копии prf3 на прерванную копию подтверждают с помощью двух последовательных гибридизаций. Так, полную ДНК полученных клонов рестриктировали с помощью различных ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом, соответствующим кластеру Ωhyg (см. выше), для подтверждения наличия кластера в геномной ДНК полученных клонов. Вторую гибридизацию осуществляли, используя в качестве зонда вставку XhoI-XhoI плазмиды pOS49.11, которая содержит четыре открытые рамки считывания (две полные и две укороченные, в которых orf3 находится целиком). Подтверждение генотипа также может быть осуществлено любым способом, известным специалисту, и, в частности, путем PCR, используя соответствующие олигонуклеотиды и секвенирование продукта PCR. Был выбран один из клонов orf3:: Ωhyg, полученный таким образом и генотип которого подтвержден, и назван OS49.16.
Продуцирование спирамицина таким образом полученным клоном OS49.16 тестировали с помощью теста на продуцирование, описанного ниже (см. пример 15). Таким образом, было показано, что этот штамм не продуцирует больше спирамицина, что подтверждает участие orf3 и/или генов, расположенных ниже, как, например, orf4, в биосинтезе спирамицинов.
Во-первых, согласно этому полученному подтверждению, секвенировали гораздо больший участок космиды pOS49.1 с той и другой стороны исследованного выше фрагмента SacI. Таким образом, исходя из космиды pOS49.1, была получена последовательность области, включающей семь полных открытых рамок считывания и две другие неполные открытые рамки считывания, расположенные с той и другой стороны этих семи полных открытых рамок считывания. За счет поиска в базах данных показано, что одна неполная из открытых рамок считывания соответствует локусу srmG (область, кодирующая фермент, называемый “поликетидсинтаза” (PKS)). Соответствующие гены были клонированы Burgett S. и др. в 1996 г. (патент США 5945320). Кроме того, в базах данных не были обнаружены другие открытые рамки считывания: семь полных открытых рамок считывания, названных orf1, orf2, orf3, orf4, orf5, orf6, orf7 (SEQ ID № 23, 25, 28, 30, 34, 36, 40), и начало восьмой открытой рамки считывания, названной orf8 (полная последовательность этой открытой рамки считывания представлена в виде SEQ ID № 43).
Пример 3
Выделение и характеристика других генов, участвующих в биосинтезе спирамицинов в Streptomyces ambofaciens
Во вторых, и с целью клонирования других генов, участвующих в биосинтезе спирамицинов, в той же самой области были выделены другие космиды, включающие фрагменты генома S. ambofaciens. Для этого осуществляли новую серию гибридизаций в колониях, используя три зонда:
- Первый используемый зонд соответствует фрагменту ДНК BamHI-PstI длиной 3,7 т.н., содержащему фрагмент гена PKS (гены, соответствующие PKS, были клонированы Burgett S. и др. в 1996 г. (патент США 5945320)), orf1, orf2 и начало orf3, субклонированному, исходя из pOS49.1, и идущему от сайта BamHI, расположенного на 1300 пар оснований выше сайта EcoRI, определяющего положение 1 последовательности SEQ ID № 1, вплоть до сайта PstI, находящегося в положении 2472 (SEQ ID № 1). Этот фрагмент BamHI-PstI субклонировали, исходя из pOS49.1, в плазмиде рВС SK+, что позволило получать плазмиду pOS49.28.
- Второй зонд соответствует фрагменту ДНК PstI-BamHI длиной около 2 т.н., содержащему фрагмент orf7 и фрагмент orf8, субклонированному, исходя из pOS49.1, и идущему от сайта PstI, находящегося в положении 6693 SEQ ID № 1, вплоть до сайта BamHI, находящегося в положении 8714 SEQ ID № 1. Этот фрагмент PstI-BamHI субклонировали, исходя из pOS49.1, в плазмиде pBC SK+, что позволило получать плазмиду pOS49.76.
- Также был использован третий зонд. Этот последний соответствует фрагменту ДНК длиной 1,8 т.н. EcoRI-HindIII, содержащему ген srmD. Ген srmD представляет собой ген, выделенный из S. ambofaciens, способный придавать резистентность к спирамицину. В самом деле, согласно предыдущим работам, осуществляли клонирование нескольких детерминант резистентности S. ambofaciens, придающих резистентность к спирамицину, в штамме S. griseofuscus (штамм, чувствительный к спирамицину) (Pernodet и др., 1993; Pernodet и др., 1999). Для выделения генов резистентности был реализован космидный банк геномных ДНК штамма S. ambofaciens АТСС23877 в космиде pKC505 (Richardson M.A. и др., 1987). Для этого, геномную ДНК штамма S.ambjfaciens АТСС23877 частично рестриктировали с помощью Sau3AI для получения фрагментов длиной от примерно 30 т.н. до 40 т.н. Таким образом рестриктированную геномную ДНК (3 мкг) лигировали с 1 мкг pKC505, предварительно рестриктированной ферментом BamHI (Pernodet и др., 1999). Смесь, полученную в результате лигирования, затем инкапсидировали in vitro в фаговые частицы. Полученные фаговые частицы были использованы для инфицирования штамма E.coli HB101 (доступный, в частности, из Американской коллекции типовых культур (АТСС) (Manassas, Virginie, США) под номером 33694). Были получены около 20000 клонов E.coli, резистентных к апрамицину, в виде пула и были проэкстрагированы космиды этих клонов. Этот пул космид вводили путем трансформации протопластов в штамм DSM 10191 S. griseofuscus (Cox K.L. & Baltz R.H., 1984), по своей природе чувствительный к спирамицину (Rao R.N. и др., 1987; этот штамм депонирован, в частности, в Немецкой коллекции микроорганизмов и клеточных культур Гмбх (DSMZ) (Брауншвейг, Германия) под номером DSM 10191). Трансформанты селекционировали на среде, содержащей апрамицин. 1300 Клонов, растущих на среде, содержащей апрамицин, переносили на среду, содержащую 5 мкг/мл спирамицина. Несколько клонов, резистентных к апрамицину, также выращивали на среде, содержащей спирамицин, и космиды этих колоний экстрагировали и использовали для трансформации E.coli S. griseofuscus (Pernodet и др., 1999). Таким образом были получены пять космид, способных придавать резистентность к апрамицину в случае E.coli и сорезистентность к апрамицину и спирамицину в случае S. griseofuscus. Среди этих 5 космид было определено, что одна космида, названная pOS44.1, в своей вставке содержит ген (SEQ ID № 15), который кодирует протеин (SEQ ID № 16), обладающий некоторым сходством с протеином, кодируемым геном mdmA Streptomyces mycofaciens (SEQ ID № 88); этот ген был назван srmD (см. сопоставление путем выравнивания, представленное на фиг.26, реализуемое по программе FASTA (cм. Pearson W.R. & Lipman D.J., 1988; Pearson W.R., 1990); доступные, в частности, из центра ресурсов INFOBIOGEN, Evry, Франция).
Для выделения детерминанты резистентности, содержащейся в плазмиде pOS44.1, ее частично рестриктировали с помощью фермента рестрикции Sau3AI для получения фрагментов размером от примерно 1,5 т.н. до 3 т.н., эти фрагменты лигировали в векторе pIJ486, линеаризированном с помощью фермента BamHI (Ward и др., 1986). Плазмиду селекционировали по ее способности придавать резистентность к спирамицину штамму DSM 10191 S. griseofuscus (Rao R.N. и др., 1987), по своей природе чувствительному к спирамицину (см. выше). Для этого пул плазмид, соответствующий фрагменту Sau3AI pOS44.1, лигированных в векторе pIJ486 (см. выше), вводили путем трансформации протопластов в штамм DSM 10191 и трансформанты селекционировали по их резистентности к тиострептону (возникающей за счет гена tsr, находящегося в pIJ486). Клоны, растущие на среде, содержащей тиострептон, переносили на среду, содержащую спирамицин. Несколько клонов, резистентных к тиострептону, также культивировали на среде, содержащей спирамицин, и экстрагировали плазмиды этих клонов. Плазмиду, придающую резистентность и содержащую вставку длиной около 1,8 т.н., отбирали и она была названа pOS44.2. Эта вставка длиной 1,8 т.н. может быть легко извлечена благодаря сайту HindIII и сайту EcoRI, находящимся в векторе с той и другой стороны вставки. Эту вставку длиной 1,8 т.н. HindIII-EcoRI секвенировали и ген резистентности, который она включает, был назван srmD. Этот фрагмент, содержащий ген smrD, таким образом, может быть легко субклонирован в векторе pUC19 (номер доступа в банк генов: М77789), рестриктированном с помощью EcoRI-HindIII; полученная плазмида была названа pOS44.4. Вставку длиной 1,8 т.н. HindIII-EcoRI, содержащую ген srmD, этой плазмиды использовали в качестве зонда для локализации генов биосинтеза спирамицина (см. выше).
Образец штамма Escherichia Coli DH5α, содержащего плазмиду pOS44.4, был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 10 июля 2002 г. под регистрационным номером I-2918.
Примерно 2000 клонов банка, полученных выше (см. пример 1), переносили на фильтр для гибридизации колоний согласно классическим способам (Sambrook и др., 1989).
Три зонда, описанных выше, метили с помощью 32Р по способу “произвольного примирования” (набор, выпускаемый фирмой Roche) и использовали для гибридизации 2000 клонов банка, перенесенных на фильтр. Гибридизацию осуществляли при температуре 65°С в буфере, описанном Church и Gilbert (Church & Gilbert,1984). Промывку осуществляли в 2Х SSC при температуре 65° в течение 15 минут и затем осуществляли две последовательные промывки в 0,5Х SSC при температуре 65°С продолжительностью 15 минут каждая. В этих условиях гибридизации и промывки 16 клонов из 2000 гибридизованных обладали сильным сигналом гибридизации по меньшей мере с одним из зондов. Однако ни одна космида не гибридизуется с тремя зондами. 16 Космид были извлечены и рестриктированы с помощью фермента рестрикции BamHI. Сравнение друг с другом профилей рестрикции этих различных космид привело к выбору двух космид, способных содержать наиболее длинные вставки области и не обладающих общими зонами. Таким образом, были выбраны две космиды, из которых одна была названа pSPM5, а другая - pSPM7. Космида pSPM5 гибридизуется с зондами prf1 - orf4 и зондом orf8, но не гибридизуется с зондом srmD. Космида pSPM7 гибридизуется только с зондом srmD и не гибридизуется с двумя другими зондами.
Эти две космиды были полностью секвенированы методом секвенирования “выстрел из дробового ружья”. Последовательности вставок этих двух космид pSPM7 и pSPM5 могут быть объединены, так как, хотя и не перекрываются, каждая из вставок включает известную последовательность на одном из своих концов. В самом деле, каждая из этих вставок включает фрагмент последовательности одного из генов, кодирующих фермент, называемый “поликетидсинтаза” (PKS). Эти 5 генов были клонированы Burgett S. и др. в 1996 г. (патент США 5945320) (см. фиг.2). Таким образом, была определена одна последовательность геномной ДНК S. ambofaciens. Последовательность из 30943 нуклеотидов, начинающаяся, в 5', с сайта EcoRI, находящегося в первом гене PKS, и идущая вплоть до сайта BamHI, в 3', представлена в виде последовательности SEQ ID № 1. Эта последовательность соответствует области выше генов PKS (см. фиг.2 и 3). Вторая область из 11171 нуклеотидов, начинающаяся с сайта PstI в 5' и идущая вплоть до сайта NcoI в 3', расположенного в пятом гене PKS, представлена в виде последовательности SEQ ID № 2. Эта вторая область представляет собой область ниже генов PKS (низ и верх определяются ориентацией 5 генов PKS, которые все имеют одну и ту же смысловую ориентацию) (см. фиг.2 и 3).
Пример 4
Анализ нуклеотидных последовательностей, определение открытых рамок считывания и характеристика генов, участвующих в биосинтезе спирамицинов
Полученные последовательности анализировали с помощью программы FramePlot (Ishikawa J. & Hotta K.,1999). Этот анализ позволяет идентифицировать, среди открытых рамок считывания, открытые рамки считывания, представляющие типичное использование кодонов Streptomyces. Этот анализ позволил определить, что эта область включает 35 открытых рамок считывания, фланкированных с той и другой стороны пятью генами, кодирующими фермент “поликетидсинтазу” (PKS). Соответственно, 10 и 25 открытых рамок считывания были индетифицированы ниже и выше этих генов (низ и верх определяются ориентацией 5 генов PKS, причем все находятся в одной и той же смысловой ориентации) (см. фиг.3). Таким образом, 25 открытых рамок считывания этого типа, занимающих область примерно 31 т.н. (SEQ ID № 1 и фиг.3), были идентифицированы выше 5 генов, кодирующих PKS, и 10 открытых рамок считывания, занимающих область примерно 11,1 т.н. (SEQ ID № 2 и фиг.3), были идентифицированы ниже генов PKS. Гены верхней области были названы: orf1, orf2, orf3, orf4, orf5, orf6, orf7, orf8, orf9c, orf10, orf11c, orf12, orf13c, orf14, orf15c, orf16, orf17, orf18, orf19, orf20, orf21c, orf22c, orf23c, orf24c et orf25c (SEQ ID № 23, 25, 28, 30, 34, 36, 40, 43, 45, 47, 49, 53, 60, 62, 64, 66, 68, 70, 72, 74, 76, 78, 80, 82 и 84). Гены нижней области были названы: orf1*c, orf2*c, orf3*c, orf4*c, orf5*, orf6*, orf7*c, orf8*, orf9*, orf10* (SEQ ID № 3, 5, 7, 9, 11, 13, 15, 17, 19 и 21).
Буква “с”, добавленная в конце названия гена, для рассматриваемой открытой рамки считывания означает, что кодирующая последовательность находится в обратной ориентации (кодирующей нитью, следовательно, является нить, комплементарная последовательности, представленной в виде SEQ ID № 1 или SEQ ID № 2 для этих генов) (см. фиг.3).
Протеиновые последовательности, следующие из этих открытых рамок считывания, сравнивали с таковыми, имеющимися в различных базах данных, с помощью разных программ: BLAST (Altschul и др., 1990; Altschul и др., 1997), CD-search, COGs (Кластер ортологичных групп) (эти три программы доступны, в частности, из Национального информационного центра по биотехнологии (NCBI) (Bethesda, Maryland, США)), FASTA ((Pearson W. R. & Lipman D. J., 1988) и (Pearson W. R., 1990)), BEATY (Worley K. C. и др., 1995) (эти две программы доступны, в частности, из центра ресурсов INFOBIOGEN, Evry, Франция)). Эти сравнения позволили сформулировать гипотезы в отношении функции вырабатываемых этими генами продуктов и идентифицировать такие, которые могут участвовать в биосинтезе спирамицина.
Пример 5
Инактивация гена: принцип конструирования прерванного штамма Streptomyces ambofaciens
Используемые способы состоят в осуществлении замены гена. Прерываемый ген-мишень заменяли копией этого гена, прерванной кластером, придающим резистентность к антибиотику (как, например, апрамицин или гигромицин), как это поясняется на фиг. 9. Используемые кластеры фланкированы с той и другой стороны кодонами терминации трансляции во всех рамках считывания и активными терминаторами транскрипции Streptomyces.
Инсерция кластера в ген-мишень может сопровождаться или нет делецией в этом гене-мишени. Размер фланкирующих областей кластера может составлять от нескольких сотен до нескольких тысяч пар оснований.
Конструкции, необходимые для инактивации гена с помощью кластера, получали в E.coli, стандартном организме для получения конструкций рекомбинантной ДНК. Прерванный ген получали в плазмиде, реплицирующейся в E.coli, но не способной реплицироваться в Streptomyces.
Конструкции затем субклонировали в векторах для возможности осуществления трансформации и инактивации желательного гена S. ambofaciens. Для этого были использованы две плазмиды:
- pOJ260 (Bierman M. и др., 1992) (см. пример 2), которая придает резистентность к апрамицину в случае E.coli и Streptomyces и которая была использована, когда ген-мишень был прерван кластером, придающим резистентность к гигромицину;
- pOSK1205 (472 пары оснований). Эта плазмида происходит от плазмиды рВК-CMV (выпускается фирмой Stratagene (LaJolla, Калифорния, США)), в которой фрагмент AvrII, содержащий последовательность, кодирующую резистентность к неомицин/канамицину, заменен последовательностью, кодирующей резистентность к гигромицину, полностью сохраняя промотор P SV40. Для этого плазмиду pHP45-Ωhyg (Blondolet-Rouault и др., 1997) рестриктировали ферментами NotI и PflmI и фрагмент, придающий резистентность к гигромицину, субклонировали на уровне сайта AvrII вектора pBK-CMV после того, как все концы были сделаны свободными концами за счет обработки ферментом Кленова. В pOSK1205, кластер, который придает резистентность к гигромицину, предшествует промотору pSV40. Эта плазмида придает резистентность к гигромицину в случае E.coli и Streptomyces и была использована, когда ген-мишень был прерван кластером, придающим резистентность к апрамицину.
Кластеры встраивали в ген-мишень либо путем клонирования, используя сайты рестрикции, имеющиеся в гене-мишени, либо путем рекомбинации между короткими идентичными последовательностями, как описывается, например, Chaveroche и др. (Chaveroche М.К.и др., 2000).
Плазмида, несущая ген, прерванный кластером, затем может быть введена в Streptomyces ambofaciens, например, путем конъюгации между E.coli и Streptomyces (Mazodier P. и др., 1989). Этот способ использовали в случае, где базовым вектором является вектор pOJ260. Может быть использован второй способ: способ трансформации протопластов после денатурации за счет щелочной обработки ДНК (Kieser T. и др., 2000) для увеличения частоты рекомбинации, как описывается, например, Oh и Chater (Oh & Chater, 1997). Этот способ был использован в случае, где базовым вектором является pOJ260 или pOSK1205 (см. ниже). Трансформанты затем селекционировали с помощью антибиотика, соответствующего кластеру, находящемуся в гене-мишени (см. фиг.9, антибиотик В). Таким образом, селекционируют смесь клонов, среди которых имела место интеграция за счет одного или двух явлений рекомбинации. Во-вторых, разыскивают клоны, чувствительные к антибиотику, в котором ген резистентности находится в векторе (вне кластера рекомбинации) (см. фиг.9, антибиотик А). Можно также отбирать клоны, в которых, в приниципе, были два явления рекомбинации, приводящие к замене исходного гена копией, прерванной кластером. Эти стадии схематически представлены на фиг.9.
Для прерывания генов-мишеней может быть использовано несколько кластеров. Можно использовать, например, кластер Ωhyg, который придает резистентность к гигромицину (Blondelet-Rouault и др., 1997; номер доступа в банк генов: Х99315).
Пример 6
Конструирование штамма Streptomyces ambofaciens, прерванного в открытой рамке считывания в гене orf3
Ген orf3 прерывали с помощью кластера Ωhyg (см. пример 2.2.) и показано, что штамм orf3::Ωhyg не продуцирует больше спирамицин, что подтверждает участие одного или нескольких генов клонированной области в биосинтезе спирамицинов (см. пример 2.2). Учитывая их ориентацию, вероятна сотранскрипция открытых рамок считывания 1-7 (см. фиг.3) и наблюдаемый фенотип (не являющийся продуцентом спирамицинов) может возникать вследствие инактивации одного или нескольких генов, подвергнутых сотранскрипции с orf3. Для подтверждения участия orf3 в биосинтезе спирамицинов осуществляли новую инактивацию гена orf3, причем эту последнюю осуществляли в открытой рамке считывания. Для этого подвергали делеции внутренний фрагмент DraIII orf3 длиной 504 пар оснований. Фрагмент ДНК, получаемый из pOS49.1, идущий от сайта EcoRI, расположенного в положении 1 (SEQ ID № 1) вплоть до сайта SacI, находящегося в положении 5274 (SEQ ID № 1), и который включает делецию между двумя сайтами DraIII в положениях 2563 и 3067 (504 удаленных нуклеотидов), клонировали в плазмиде pOJ260 (Bierman M. и др., 1992). Таким образом полученная плазмида была названа pOS49.67.
Вставка pOS49.67, следовательно, образована фрагментом ДНК S. ambofaciens, содержащим гены orf1, orf2, orf3 с делецией в рамке, orf4 и часть orf5. Вектором, в котором была субклонирована эта вставка, является pOJ260; плазмида pOS49.67, следовательно, придает резистентность к апрамицину и была введена путем трансформации протопластов в штамм OS49.16 (см. пример 2). Были получены штамм OS49.16, обладающий резистентностью к гигромицину, трансформанты hygR и apraR. После двух пассажей таких клонов на неселективной среде были обнаружены клоны, чувствительные к апрамицину и гигромицину (apraS и hygS). В некоторых из этих клонов, на деле, ожидают, что явление рекомбинации между гомологичными последовательностями приводит к замене копии orf3, прерванной кластером Ωhyg (содержащимся в геноме штамма OS49.16), копией orf3 с делецией в рамке считывания, находящейся в векторе. Клоны, полученные в результате этой рекомбинации, ожидаются как клоны apraS и hygS после элиминации последовательностей вектора. Генотип таким образом полученных штаммов может быть подтвержден путем гибридизации или путем PCR и секвенирования продукта PCR (для подтверждения того, что одна копия orf3 с делецией в рамке считывания имеется в геноме полученных клонов). Таким образом, были получены клоны, обладающие только копией orf3 с делецией в рамке считывания, и их генотип был подтвержден. В особенности был отобран клон, обладающий искомыми характеристиками, и назван как OS49.67.
Образец штамма OS49.67 был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 10 июля 2002 г. под регистрационным номером I-2916.
Пример 7
Конструирование штамма Streptomyces ambofaciens, прерванного в гене orf8
Для осуществления инактивации гена orf8 получали конструкцию, в которой кластер Ωhyg был введен в последовательность, кодирующую orf8. Для этого сначала конструировали плазмиду pOS49.88. Плазмида pOS49.88 производится от плазмиды pUC19 (номер доступа в банк генов: М77789) за счет инсерции фрагмента длиной 3,7 т.н. (фрагмент PstI-EcoRI, полученный из космиды pSPM5), содержащего конец orf7, orf8 и начало orf9, клонированного в сайтах PstI-EcoRI pUC19. Кластер Ωyg (в виде фрагмента BamHI со свободными концами за счет обработки ферментом Кленова) был клонирован на уровне одного сайта SalI pOS49.88, расположенного в orf8, после того, как все концы были сделаны свободными концами путем обработки ферментом Кленова.
После клонирования с получением свободных концов, были получены два типа плазмид согласно направлению инсерции кластера: pOS49.106, где гены hyg и orf8 находятся в одинаковой ориентации, и pOS49.120, где гены hyg и orf8 находятся в противоположных ориентациях. Вставку плазмиды pOS49.106 затем субклонировали в плазмиде pOJ260 для получения pOS49.107. Для этого плазмиду pOS49.106 рестриктировали с помощью фермента Asp7181 и концы делали свободными концами путем обработки ферментом Кленова, этот продукт рестрикции повторно рестриктировали с помощью фермента PstI и фрагмент, содержащий ген orf8, в который встроен кластер Ωhyg, клонировали в векторе pOJ260 (см. выше). Для этого вектор pOJ260 рестриктировали ферментами EcoRI и PstI и использовали для лигирования. Эта манипуляция позволяет, следовательно, достигать направленного лигирования, поскольку каждый из обоих фрагментов имеет свободный конец с одной стороны и PstI с другой стороны. Полученная плазмида была названа pOS49.107.
Плазмида pOS49.107 была введена в штамм АТСС23877 S.ambofaciens путем трансформации протопластов (Kieser T. и др., 2000). После трансформации протопластов клоны селекционировали по их резистентности к гигромицину. Резистентные к гигромицину клоны затем пересевали, соответственно, на среду с гигромицином (антибиотик В) и среду с апрамицином (антибиотик А) (см. фиг.9). Клоны, резистентные к гигромицину (HygR) и чувствительные к апрамицину (ApraS), в принципе, являются такими, где осуществлен двойной кроссинговер и которые обладают геном orf8, прерванным кластером Ωhyg. Замена исходной копии orf8 на копию, прерванную кластером Ωhyg, была подтверждена путем саузерн-блоттинга. Таким образом, полную ДНК полученных клонов рестриктировали с помощью нескольких ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом, соответствующим кластеру Ωhyg, для подтверждения наличия кластера в геномной ДНК полученных клонов. Вторую гибридизацию осуществляли при использовании в качестве зонда вставки PstI-EcoRI, содержащей конец orf7, orf8 и начало orf9, размером около 3,7 т.н. плазмиды pOS49.88. Подтверждение генотипа может быть осуществлено любым способом, известным специалисту, и, в частности, путем PCR, используя соответствующие олигонуклеотиды, и секвенироания продукта РСR.
Был отобран клон orf8:: Ωhyg и назван OS49.107. Образец штамма OS49.107 был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 10 июля 2002 г. под регистрационным номером I-2917.
Пример 8
Конструирование штамма Streptomyces ambofaciens, прерванного в гене orf10
Фрагмент ДНК длиной 1,5 т.н., внутренний по отношению к гену orf10, получали путем PCR при использовании в качестве матрицы геномной ДНК S. ambofaciens и следующих праймеров:
SRMR1: 5' CTGCCAGTCCTCTCCCAGCAGTACG 3' (SEQ ID № 89)
SRMR2: 5' TGAAGCTGGACGTCTCCTACGTCGG 3' (SEQ ID № 90).
Этот фрагмент ДНК, получаемый в результате PCR, клонировали в векторе pCR2.1, выпускаемом фирмой Invitrogen (Карлсбад, Калифорния, США). Таким образом полученная плазмида была названа pOS49.32. Кластер Ωhyg (в виде фрагмента BamHI, см. выше) клонировали на уровне одного сайта BstEII, внутреннего по отношению к фрагменту гена orf10, после того, как все концы делали свободными концами за счет обработки ферментом Кленова. После клонирования с получением свободных концов, получали два типа плазмид согласно направлению инсерции кластера: pOS49.43, где гены hyg и orf10 находятся в одинаковой ориентации, и pOS49.44, где гены hyg и orf10 находятся в противоположных ориентациях. Вставку плазмиды pOS49.43 переносили (в виде фрагмента Asp718I-XbaI, концы которого были высвобождены путем обработки ферментом Кленова) в сайт EcoRI плазмиды pOJ260, что позволило получить плазмиду pOS49.50. Плазмиду pOS49.50, содержащую фрагмент гена orf10, прерванного кластером Ωhyg, вводили в штамм АТСС23877 Streptomyces ambofaciens. После трансформации клоны селекционировали по их резистентности к гигромицину. Резистнтные к гигромицину клоны затем пересевали, соответственно, на среду с гигромицином (антибиотик В) и на среду с апрамицином (антибиотик А) (см. фиг.9). Клоны, резистентные к гигромицину (HygR) и чувствительные к апрамицину (ApraS), в принципе, являются такими, где осуществлен двойной кроссинговер и которые обладают геном orf10, прерванным кластером Ωhyg. Таким образом были получены клоны, которые обладали маркером резистентности к гигромицину, находящимся в кластере, и которые утратили маркер резистентности к апрамицину, находящийся в векторе pOJ260. Замена исходной копии orf10 на копию, прерванную кластером orf10::Ωhyg, была подтверждена путем саузерн-блоттинга. Таким образом, полную геномную ДНК полученных клонов рестриктировали с помощью нескольких ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом, соответствующим кластеру Ωhyg, для подтверждения наличия кластера в геномной ДНК полученных клонов. Вторую гибридизацию осуществляли при использовании в качестве зонда продукта PCR длиной 1,5 т.н., внутреннего по отношению к гену orf10 (см. выше).
В особенности был отобран клон, имеющий ожидаемые характеристики (orf10::Ωhyg), и назван OS49.50. На самом деле, путем двух гибридизаций было подтверждено, что кластер Ωhyg находится в геноме этого клона и что в самом деле достигают ожидаемого профиля рестрикции в случае замены, вследствие двойного явления рекомбинации, исходного гена на копию, прерванную кластером Ωhyg в геноме этого клона. Подтверждение генотипа также может быть осуществлено любым, известным специалисту способом, и, в частности, путем PCR, используя соответствующие олигонуклеотиды, и секвенирования продукта PCR.
Пример 9
Инактивация генов: принцип конструирования штамма Streptomyces ambofaciens, прерванного по методу “эксцизируемых кластеров” (см. фиг.9 и 10)
Для инактивации генов может быть использован второй тип кластеров: кластеры, называемые “эксцизируемыми кластерами”. Эти кластеры обладают преимуществом в том, что могут быть эксцизированы из Streptomyces путем сайтспецифической рекомбинации, после того, как были введены в геном S. ambofaciens. Целью является инактивация некоторых генов в штаммах Streptomyces без оставления в конечном штамме маркеров селекции или больших участков ДНК, не относящихся к штамму. После эксцизии существует только короткая последовательность длиной примерно тридцать пар оснований (называемая “рубцовым” сайтом) в геноме штамма (см. фиг.10).
Осуществление этой системы состоит, во-первых, в замене исходной копии гена-мишени (за счет двух явлений гомологичной рекомбинации, см. фиг.9) конструкцией, в которой эксцизируемый кластер встроен в этот ген-мишень. Инсерция этого кластера сопровождается делецией в гене-мишени (см. фиг.9). Во-вторых, провоцируется эксцизия эксцизируемого кластера из генома штамма. Эксцизируемый кластер функционирует благодаря системе сайтспецифической рекомбинации и обладает преимуществом в том, что позволяет получать мутанты Streptomyces, не несущие в конечном счете гена резистентности. Также избавляются от возможных полярных воздействий на экспрессию генов, расположенных ниже инактивированного гена или инактивированных генов (см. фиг.10).
Использование эксцизируемых кластеров описано и применено в случае многочисленных организмов, в том числе в случае клеток млекопитающих, дрожжевых клеток и в E.coli (Bayley и др., 1992; Brunelli и Pall, 1993; Camilli и др., 1994; Dale и Ow, 1991; Russel и др., 1992; Lakso и др., 1992). Эти эксцизируемые кластеры, все, используют сайтспецифическую рекомбиназу Cre, воздействующую на сайты lox. Эта система рекомбинации происходит от бактериофага Р1.
Для конструкции системы типа “эксцизируемый кластер” в Streptomyces, извлекали часть системы сайтспецифической рекомбинации, описанной для мобильного генетического элемента pSAM2 Streptomyces ambofaciens (Boccard и др., 1989 а и b). Создание системы состоит, во-первых, в конструировании рекомбинантного вектора, включающего прерываемый ген, в который встроен эксцизируемый кластер. Инсерция в ген-мишень эксцизируемого кластера сопровождается делецией в гене-мишени. Она может быть реализована путем клонирования при использовании сайтов рестрикции, имеющихся в гене-мишени, или путем рекомбинации между короткими идентичными последовательностями, как описывается, например, Chaveroche и др. (Chaveroche M.K. и др., 2000). Эксцизируемый кластер может быть сконструирован при использовании, например, кластера Ωhyg (Blondelet-Rouault и др., 1997). Этот кластер фланкирован последовательностями attR и attL, которые обычно фланкируют интегрированную копию pSAM2 (см. фиг.15). Последовательности attR и attL, все, содержат сайты, необходимые для сайтспецифической рекомбинации, позволяющей осуществлять эксцизию pSAM2 или любого фрагмента ДНК, расположенного между этими двумя областями (Sezonov и др., 1997; Raynal и др., 1998). Конструкция такого кластера, конечно, не ограничена использованием кластера Ωhyg, но и другие кластеры резистентностей могут служить базой для конструкции этого кластера (например, кластер Ωaac или Ωvph (Blondelet-Rouault и др., 1997).
После получения этой конструкции штамм Streptomyces трансформировали с помощью рекомбинантной плазмиды. Трансформанты затем селекционировали с антибиотиком, соответствующим кластеру, находящемуся в гене-мишени (см. фиг.9, антибиотик В; речь идет, например, о селекции с помощью гигромицина, если эксцизируемый кластер происходит от кластера Ωhyg). Таким образом, селекционируют смесь клонов, среди которых имела место интеграция за счет одного или за счет двух явлений рекомбинации. Во-вторых, разыскивают клоны, чувствительные к антибиотику, в случае которых ген резистентности находится в векторе (вне кластера рекомбинации) (см. фиг.9, антибиотик А). Можно также селекционировать клоны, в которых имели место, в принципе, два явления рекомбинации, приводящих к замене исходного гена копией, прерванной кластером. Эти стадии схематически представлены на фиг.9; генотип таким образом полученных клонов подтвержден саузерн-блоттингом и отобран штамм, обладающий желательными характеристиками (замена исходного гена копией, прерванной эксцизируемым кластером).
Во-вторых, штамм, отобранный выше, трансформировали с помощью плазмиды, позволяющей осуществлять экспрессию генов xis и int, которые оба необходимы для сайтспецифической рекомбинации между сайтами attR и attL. Следствием этой рекомбинации является удаление эксцизируемого кластера из генома штамма за счет явления рекомбинации (см. фиг.10) (Raynal и др., 1998). Представляет интерес выбор вектора, несущего гены xis и int, из относительно нестабильных векторов у Streptomyces (например, производное вектора Streptomyces pWHM3 (Vara и др., 1989)), это позволяет получать штамм, утративший этот последний вектор после нескольких циклов споруляции в отсутствие давления отбора.
Для эксцизии кластера можно, например, использовать плазмиду pOSV508 (см. фиг.14), которая введена путем трансформации протопластов в штамм S. ambofaciens, содержащий ген, прерванный эксцизируемым кластером. Плазмида pOSV508 происходит от плазмиды pWHM3 (Vara J. и др., 1989) (см. фиг.13), в которую добавлены гены xis и int pSAM2 (Boccard F. и др., 1989 b), находящиеся под контролем промотора ptrc (Amann E. и др., 1988). Гены xis и int, находящиеся под контролем промотора ptrc, были субклонированы в плазмиде pWHM3, исходя из плазмиды pOSint3 (Raynal и др., 1998) (см. фиг.14). Введение в мутантный штамм плазмиды pOSV508, несущей гены xis и int pSAM2, позволяет осуществлять эффективную эксцизию путем сайтспецифической рекомбинации эксцизируемого кластера между сайтами attL и attR, фланкирующими этот кластер (Raynal A. и др., 1998) (фиг.10). Среди трансформантов, селекционированных по их резистентности к тиострептону, вызванной геном tsr, который несет pOSV508, выбирают такие, которые стали чувствительными к антибиотику, присутствие которого в кластере придает резистентность (см. фиг.10). Эксцизия является эффективной и обнаружено, что более 90% трансформатов относятся к этому типу. После одного или нескольких циклов роста и споруляции на твердой среде, лишенной тиострептона, получают клоны, утратившие плазмиду pOSV508. Эти клоны улавливаются по их чувствительности к тиострептону. Последовательность гена-мишени, подвергнутого делеции, может быть подтверждена с помощью PCR и секвенирования продукта PCR.
Наконец, полученный в результате штамм на уровне инактивированного гена (например, внутренняя делеция) включает “рубцовый” сайт att, соответствующий минимальному сайту attB (Raynal и др., 1998), который происходит от рекомбинации между сайтами attR и attL. Этот минимальный сайт attB, который продолжает существовать, подобен таковому, присутствующему естественным образом в штаммах Streptomyces ambofaciens, Streptomyces pristinaespiralis и Streptomyces lividans (Sezonov и др., 1997).
Ген, который хотят инактивировать, может быть сотранскрибирован с другими генами, расположенными ниже. Для избежания того, что инактивация одного из генов оказывает полярное воздействие на экспрессию генов, находящихся ниже, в опероне, важным является достижение делеции в рамке считывания после эксцизии кластера. Система эксцизируемых кластеров, такая, как описанная выше, позволяет отвечать такому требованию. В самом деле, специалист может легко сконструировать три различных эксцизируемых кластера, оставляющие после эксцизии последовательность из 33, 34 или 35 нуклеотидов, соответственно, без стоп-кодона, какая бы ни была рамка считывания. Зная последовательность гена-мишени и размер делеции, ассоциированной с инсерцией кластера, можно выбирать между этими тремя эксцизируемыми кластерами таким образом, чтобы эксцизия приводила к делеции в рамке считывания. На 33, 34 или 35 добавленных нуклеотидов 26 соответствуют миимальной последовательности attB (см. фиг.27).
В случае настоящей заявки были использованы два эксцизируемых кластера. Эти два кластера представляют собой следующие: att1Ωhyg+ (Seq ID № 91) и att3Ωaac- (SEQ ID № 92); эти кластеры после эксцизии оставляют, соответственно, 33 и 35 нуклеотидов. Они включают, соответственно, кластер Ωhyg или кластер Ωaac, причем знаки + и - соответствуют ориентации кластера резистентности. Эти два кластера были сконструированы и клонированы на уровне сайта EcoRV вектора pBC SK+, сайт которого HindIII предварительно был ликвидирован. Полученные плазмиды были названы patt1Ωhyg+ и patt3Ωaac-, соответственно. Эксцизируемые кластеры могут быть легко устранены путем рестрикции плазмиды с помощью EcoRV.
Пример 10
Конструирование штамма Streptomyces ambofaciens, прерванного в гене orf2
Инактивацию гена orf2 реализовали по способу с использованием эксцизируемых кластеров (см. выше). Используемым исходным штаммом является Streptomyces ambofaciens OSC2, который происходит от штамма АТСС23877. Однако, штамм OSC2 отличается от штамма АТСС23877 тем, что он утратил мобильный генетический элемент pSAM2 (Boccard и др., 1989 а и b). Этот мобильный элемент может быть утрачен спонтанно в процессе протопластизации (воздействие лизоцима для рестрикции бактериальной стенки и фрагментации мицелия (Kieser и др., 2000)) и регенерации протопластов штамма АТСС23877. Для селекционирования клонов, утративших элемент pSAM2, осуществляли просеивание, основанное на репрессии гена pra с помощью репрессора транскрипции KorSA (Sezonov и др.,1995; Sezonov G. и др., 2000). Для этого фрагмент ДНК, содержащий промотор гена pra, расположенный выше гена aph (придающий резистентность к канамицину и лишенный своего собственного промотора), клонировали в нестабильном векторе pWHM3Hyg, причем этот последний происходит от плазмиды pWHM3 (Vara и др., 1989), в котором ген tsr был заменен геном hyg (придающий резистентность к гигромицину). Таким образом полученная плазмида была названа pOSV510. Штамм АТСС23877 трансформировали, после протопластизации, с помощью плазмиды pOSV510. Промотор Pra представляет собой промотор, подавляемый репрессором KorSA, причем ген, кодирующий этот последний, расположен в мобильном элементе pSAM2 (Sezonov G. и др., 2000). После трансформации с помощью плазмиды pOSV510, трансформированные бактерии селекционировали по их резистентности к канамицину (вызываемой геном aph, находящимся в pOSV510). Клоны, утратившие интегративный элемент pSAM2, утратили репрессор KorSA и промотор Pra, следовательно, более не подавлен и позволяет осуществлять экспрессию гена резистентности к канамицину aph. Селекция за счет канамицина после трансформации с помощью плазмиды pOSV510, следовательно, позволяет отбирать клоны, утратившие интегративный элемент pSAM2 (и, следовательно, KorSA) и содержащие плазмиду pOSV510. Так как плазмида pOSV510 является нестабильной после нескольких циклов споруляции без антибиотика, выделенные клоны пересевали на среду с канамицином, на среду с гигромицином и на среду без антибиотика. Клоны, чувствительные к канамицину и к гигромицину, утратили pOSV510. Потеря элемента pSAM2 была подтверждена путем гибридизации и PCR. Был отобран один клон, имеющий желательные характеристики, и назван OSC2.
Образец штамма OSC2 был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 10 июля 2002 г. под регистрационным номером I-2908.
Инактивацию гена orf2 осуществляли по способу с эксцизируемыми кластерами (см. выше и фиг.10). Для этого вставку длиной 4,5 т.н., последовательность которой идет от сайта EcoRI, расположенного в положении 1, вплоть до сайта BamHI, расположенного в положении 4521 (SEQ ID № 1), субклонировали на уровне сайтов EcoRI и BamHI плазмиды pUC19 (номер доступа в банк генов: М77789), исходя из космиды pSPM5. Таким образом полученная плазмида была названа pOS49.99.
Эта плазмида была введена в штамм E.coli KS272, который уже содержал плазмиду pKOBEG (Chaveroche и др., 2000) (см. фиг.12).
Параллельно, эксцизируемый кластер att3Ωaac- (SEQ ID № 92, см. выше) амплифицировали путем PCR при использовании в качестве матрицы плазмиды pOSK1102 (плазмида pOSK1102 представляет собой плазмиду, происходящую от вектора pGP704Not (Chaveroche и др., 2000; Miller V.L. & Mekalanos J.J., 1988), в которой кластер att3Ωaac- был клонирован как фрагмент EcoRV в одном сайте EcoRV pGP704Not) и с помощью следующих праймеров:
ORF2A
5' CCCGCGCGGCAGCCTCTCCGTGATCGAGTCCGGCGTGACCATCGCGCGCGCTTCGTTCGG-3' (SEQ ID № 93);
ORF2B
5' GCTCCGTGCGTCATGCAGGAAGGTGTCGTAGTCGCGGTAGATCTGCCTCTTCGTCCCGAA-3' (SEQ ID № 94).
40 Дезоксинуклеотидов, расположенных в конце 5' этих олигонуклеотидов, включают последовательность, соответствующую последовательности в гене-мишени (в настоящем случае orf2), и 20 дезоксинуклеотидов, расположенных ближе к 3' (представлено жирным шрифтом выше), соответствуют последовательности одного из концов эксцизируемого кластера att3Ωaac- (см. фиг.11).
Таким образом полученный продукт PCR использовали для трансформации штамма E.coli, содержащего плазмиды pKOBEG и pOS49.99, как описывает Chaveroche и др. (2000) (см. фиг.12). Таким образом, бактерии были транстформированы путем электропорации и селекционированы по их резистентности к апрамицину. Плазмиды полученных клонов были экстрагированы и рестриктированы с помощью нескольких ферментов рестрикции с целью подтверждения того, что профиль достигнутой рестрикции соответствует ожидаемому профилю, если имела место инсерция кластера (attt3Ωaac-) в ген-мишень (orf2), то есть, если действительно произошла гомологичная рекомбинация между концами продукта PCR и гена-мишени (Chaveroche и др., 2000). Подтверждение конструкции также может быть осуществлено любым способом, известным специалисту, и, в частносити, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR. Был отобран клон, плазмида которого обладает ожидаемым профилем, и соответствующая плазмида была названа pSPM17. Эта плазмида происходит от pOS49.99, в которой orf2 прерван с помощью апрамицинового кластера (см. фиг.12).
Инсерция кластера сопровождается делецией в orf2, между нуклеотидами 211 и 492 кодирующей части orf2.
Плазмиду pSPM17 рестриктировали с помощью фермента EcoRI, затем концы делали свободными концами путем обработки ферментом Кленова, этот продукт рестрикции затем рестриктировали с помощью фермента XbaI и вставку, содержащую утраченный ген orf2, клонировали в векторе pOSK1205 (см. выше). Для этого вектор pOSK1205 рестриктировали ферментом BamHI, затем концы делали свободными концами путем обработки ферментом Кленова, этот продукт после этого рестриктировали ферментом XbaI и использовали для лигирования со вставкой, полученной из pSPM17, как описывается выше. Эта манипуляция, следовательно, позволяет достигать направленного лигирования, поскольку каждый из двух фрагментов имеет свободный конец с одной стороны и XbaI с другой стороны. Таким образом полученная плазмида была названа pSPM21, она несет ген резистентности к гигромицину (векторная часть) и вставку, в которой утраченный ген orf2 заменен кластером att3Ωaac-.
Вектор pSPM21 был введен в штамм Streptomyces ambofaciens OSC2 (см. выше) путем трансформации протопластов (Kieser T. и др., 2000). После трансформации клоны селекционировали по их резистентности к апрамицину. Резистентные к апрамицину клоны затем пересевали, соответственно, на среду с апрамицином (антибиотик В) и на среду с гигромицином (антибиотик А) (см. фиг.9). Клонами, резистентными к апрамицину (ApraR) и чувствительными к гигромицину (HygS), являются, в принципе, такие, где осуществлен двойной кроссинговер и которые обладают геном orf2, прерванным кластером att3Ωaac-. Эти клоны селекционировали и подтверждали замену исходной копии orf2 копией, прерванной кластером. Наличие кластера attΩaac- было подтверждено путем PCR в колонии. Также была осуществлена гибридизация. Для этого полную ДНК полученных клонов рестриктировали с помощью нескольких ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом длиной 3 т.н., соответствующим фрагменту EcoRI-BamHI вставки плазмиды pOS49.99 (см. выше). Подтверждение генотипа также может быть осуществлено любым способом, известным специалисту, и, в частности, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR.
Был отобран клон, обладающий ожидаемыми характеристиками, и назван SPM21. Путем PCR и гибридизации может быть подтверждено, что кластер att3Ωaac- действительно находится в геноме этого клона и что в самом деле достигнут профиль рестрикции, ожидаемый в случае замены, вследствие двойного явления рекомбинации, исходного гена копией, прерванной кластером att3Ωaac-, в геноме этого клона. Этот клон, следовательно, имеет генотип: orf2:: att3Ωaac-.
Образец штамма SPM21 был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 10 июля 2002 г. под регистрационным номером I-2914.
Штамм SPM21 трансформировали с помощью вектора POSV508 за счет трансформации протопластов с целью провоцирования эксцизии кластера (см. фиг.14). Плазмида pOSV508 происходит от плазмиды pWHM3 (Vara J. и др., 1989) (см. фиг.13), в которую добавлены гены xis и int pSAM2 (Boccard F. и др., 1989 b), находящиеся под контролем промотора ptrc (Amann E. и др., 1988) (см. фиг.14). Введение в штамм SPM21 плазмиды pOSV508, несущей гены xis и int pSAM2, позволяет осуществлять эффективную эксцизию путем сайтспецифической рекомбинации эксцизируемого кластера между сайтами attL и attR, фланкирующими этот кластер (Raynal A. и др., 1998) (фиг.10). Среди трансформантов, селекционированных по их резистентности к тиострептону, вызываемой геном tsr, который находится в pOSV508, выбирают два, ставшие чувствительными к апрамицину, ген резистентности которых находится в кластере att3Ωaac-; следствием эксцизии, на деле, является утрата этого гена резистентности (см. фиг.10). Плазмида pOSV508 является нестабильной и после двух последовательных пассажей на среде без антибиотика выделенные клоны пересевают на среду с тиострептоном и на среду без тиострептона. Клоны, чувствительные к тиострептону, утратили pOSV508. То, что эксцизия кластера действительно приводит к делеции в рамке считывания в гене orf2, подтверждено с помощью PCR и секвенирования продукта PCR; в самом деле, после эксцизии кластера остается характерная “рубцовая” последовательность att3 (которая подобна сайту attB, образующемуся после рекомбинации сайтов attL и attR):
5' ATCGCGCGCGCTTCGTTCGGGACGAAGAGGTAGAT 3' (SEQ ID № 95).
Таким образом полученный и обладающий желательным генотипом (orf2::att3) штамм был назван SPM22.
Образец штамма SPM22 был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 10 июля 2002 г. под регистрационным номером I-2915.
Пример 11
Конструирование штамма Streptomyces ambofaciens, прерванного в гене orf12
Для инактивации orf12, orf13c и orf14 использовали одну и ту же исходную плазмиду (pSPM504) для введения в различные положения кластера типа “эксцизируемого кластера”. Эта плазмида имеет вставку длиной 15,1 т.н., которая соответствует области от orf7 до orf17. Для конструирования этой плазмиды фрагмент BglII длиной 15,1 т.н., происходящий от рестрикции космиды pSPM7 (см. выше), клонировали в плазмиде pMBL18 (Nakano и др., 1995), рестриктированной с помощью BamHI. Так как концы BamHI и BglII являются совместимыми, после лигирования получают плазмиду pSPM502. Всю в целом вставку pSPM502 затем субклонировали (в виде фрагмента HindIII/NheI) в плазмиде pOSK1205 (рестриктированной с помощью HindIII/NheI), что позволило получить плазмиду pSPM504.
Эта плазмида была введена в штамм E.coli KS272, который уже содержал плазмиду pKOBEG (Chaveroche и др., 2000) (см. фиг.12).
Параллельно эксцизируемый кластер att3Ωaac- амплифицировали путем PCR, используя в качестве матрицы плазмиду pOSK1102 (плазмида pOSK1102 представляет собой плазмиду, происходящую от вектора pGP704Not (Chaveroche и др., 2000; Miller V.L. & Mekalanos J.J., 1988), в котором был клонирован кластер att3Ωaac- в качестве фрагмента EcoRV в одном сайте EcoRV pGP704Not); используемыми праймерами являются следующие:
EDR8:5' CGGGATGATCGCTTGTCCGGCGGCCGGATGCCTAGCCTCATCGCGCGCGCTTCGTTCGG 3' (SEQ ID № 96);
EDR9:5' CCCGATCCAGAACGTCTGGTCGGTGATCAGGTCGCTGTTCATCTGCCTCTTCGTCCCGAA 3' (SEQ ID № 97).
40 (только 39 в EDR8) Дезоксинуклеотидов, находящихся в конце 5' этих олигонуклеотидов, включают последовательность, соответствующую последовательности в гене-мишени (в настоящем случае orf12), и 20 дезоксинуклеотидов, расположенных больше всего в 3' (представлено жирным шрифтом выше), соответствуют последовательности одного из концов эксцизируемого кластера att3Ωaac- (см. фиг.11).
Таким образом полученный продукт PCR был использован для трансформации штамма E.coli KS272, содержащего плазмиды pKOBEG и pSPM504 (см. выше), как описывается Chaveroche и др. (Chaveroche и др., 2000) (см. фиг.12 по принципу: плазмида pOS49.99 должна быть заменена плазмидой pSPM504 и полученная плазмида не является более pSPM17, а представляет собой плазмиду pSPM507). Таким образом, бактерии были трансформированы путем элктропорации с помощью этого продукта PCR и клоны были селекционированы по их резистентности к апрамицину. Плазмиды полученных клонов были экстрагированы и рестриктированы с помощью нескольких ферментов рестрикции с целью подтверждения того, что полученный профиль рестрикции соответствует ожидаемому профилю, если имела место инсерция кластера (att3Ωaac-) в ген-мишень (orf12), то есть, если действительно произошла гомологичная рекомбинация между концами продукта PCR и гена-мишени (Chaveroche и др., 2000). Подтверждение конструкции также может быть осуществлено любым способом, известным специалисту, и, в частности, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR. Клон, плазмида которого обладает ожидаемым профилем, был отобран и соответствующая плазмида была названа pSPM507. Эта плазмида происходит от pSPM504, в которой ген orf12 прерван кластером att3Ωaac- (см. фиг.12). Инсерция кластера сопровождается делецией в гене orf12, прерывание начинается на уровне тридцатого кодона orf12. После кластера остаются 46 последних кодонов гена orf12.
Вектор pSPM507 вводили в штамм Streptomyces ambofaciens OSC2 (см. выше) путем трансформации протопластов (Kieser T. и др., 2000). После трансформации клоны селекционировали по их резистентности к апрамицину. Резистентные к апрамицину клоны затем пересевали, соответственно, на среду с апрамицином (антибиотик В) и на среду с гигромицином (антибиотик А) (см. фиг.9). Клоны, резистентные к апрамицину (ApraR) и чувствительные к гигромицину (HygS), в принципе, представляют собой такие, где имел место двойной кроссинговер и которые обладают геном orf12, прерванным кластером att3Ωaac-. Эти клоны предпочтительно были селекционированы и замена исходной копии гена orf12 копией, прерванной кластером, была подтверждена путем гибридизации. Так, полную ДНК полученных клонов рестриктировали с помощью нескольких ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом, соответствующим кластеру att3Ωaac-, для подтверждения наличия кластера в геномной ДНК полученных клонов. Вторую гибридизацию осуществляли при использовании в качестве зонда фрагмента ДНК, полученного путем PCR и соответствующего очень большой части кодирующей последовательности гена orf12.
Подтверждение генотипа также может быть существлено любым способом, известным специалисту, и, в частности, путем PCR, используя соответствующие олигонуклеотиды, и секвенирования продукта PCR.
Предпочтительно был отобран клон, обладающий ожидаемыми характеристиками (orf12:: att3Ωaac-), и назван SPM507. На деле, путем двух гибридизаций было подтверждено, что кластер att3Ωaac- действительно находится в геноме этого клона и что в самом деле достигают ожидаемого профиля рестрикции в случае замены, вследствие двойной рекомбинации, исходного гена копией, прерванной кластером att3Ωaac-, в геноме этого клона. Этот клон, следовательно, имеет генотип orf12:: att3Ωaac- и был назван SPM507. Ненужным является осуществление эксцизии кластера для исследования эффекта инактивации orf12 с точки зрения ориентации генов (см. фиг.3). В самом деле, тот факт, что orf13c ориентирован в направлении, противоположном гену orf12, показывает, что эти гены не являются сотранскрибированными. Использование эксцизируемого кластера, взамен, позволяет иметь возможность в любое время избавиться от маркера селекции, в частности, путем трансформации с помощью плазмиды pOSV508.
Образец штамма SPM507 был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 10 июля 2002 г. под регистрационным номером I-2911.
Пример 12
Конструирование штамма Streptomyces ambofaciens, прерванного в гене orf13c
Эксцизируемый кластер att3Ωaac- амплифицировали путем PCR, используя в качестве матрицы плазмиду pOSK1102 (см. выше), с помощью следующих праймеров:
EDR3: 5' ACCGGGGCGGTCCTCCCCTCCGGGGCGTCACGGCCGCGGAATCTGCCTCTTCGTCCCGAA 3' (SEQ ID № 98);
EDR4: 5' CACGCAGCGAGCCGACGCACTGATGGACGACACGATGGCCATCGCGCGCGCTTCGTTCGG 3' (SEQ ID № 99).
40 Дезоксинуклеотидов, находящихся в конце 5' этих олигонуклеотидов, включают последовательность, соответствующую последовательности в гене-мишени (в настоящем случае orf13c), и 20 дезоксинуклеотидов, расположенных больше всего в 3' (представлено жирным шрифтом выше), соответствуют последовательности одного из концов эксцизируемого кластера att3Ωaac- (см. фиг.11).
Таким образом полученный продукт PCR был использован для трансформации штамма E.coli KS272, содержащего плазмиды pKOBEG и pSPM504 (см. выше), как описывается Chaveroche и др. (Chaveroche и др., 2000) (см. фиг.12 по принципу: плазмида pOS49.99 должна быть заменена плазмидой pSPM504 и полученная плазмида не является более pSPM17, а представляет собой плазмиду pSPM508). Таким образом, бактерии были трансформированы путем электропорации с помощью продукта PCR и клоны были селекционированы по их резистентности к апрамицину. Плазмиды полученных клонов были экстрагированы и рестриктированы с помощью нескольких ферментов рестрикции с целью подтверждения того, что полученный профиль рестрикции соответствует ожидаемому профилю, если имела место инсерция кластера (att3Ωaac-) в ген-мишень (orf13c), то есть, если действительно произошла гомологичная рекомбинация между концами продукта PCR и гена-мишени (Chaveroche и др., 2000). Подтверждение конструкции также может быть осуществлено любым способом, известным специалисту, и, в частности, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR. Клон, плазмида которого обладает ожидаемым профилем, был отобран и соответствующая плазмида была названа pSPM508. Эта плазмида происходит от pSPM504, в которой ген orf13c прерван апрамициновым кластером (см. фиг.12). Инсерция кластера сопровождается делецией в гене orf13c, прерывание начинается на уровне шестого кодона orf13c. После кластера остаются 3 последних кодона гена orf13c.
Вектор pSPM508 вводили в штамм Streptomyces ambofaciens OSC2 (см. выше) путем трансформации протопластов (Kieser T. и др., 2000). После трансформации клоны селекционировали по их резистентности к апрамицину. Резистентные к апрамицину клоны затем пересевали, соответственно, на среду с апрамицином (антибиотик В) и на среду с гигромицином (антибиотик А) (см. фиг.9). Клоны, резистентные к апрамицину (ApraR) и чувствительные к гигромицину (HygS), в принципе, представляют собой такие, где имел место двойной кроссинговер и которые обладают геном orf13c, прерванным кластером att3Ωaac-. Эти клоны предпочтительно были селекционированы и замена исходной копии гена orf13c копией, прерванной кластером, была подтверждена путем гибридизации. Так, полную ДНК полученных клонов рестриктировали с помощью нескольких ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом, соответствующим кластеру att3Ωaac-, для подтверждения наличия кластера в геномной ДНК полученных клонов. Вторую гибридизацию осуществляли при использовании в качестве зонда продукта PCR, соответствующего последовательности, выступающей на сотню пар оснований выше и ниже кодирующей последовательности orf13c. Подтверждение генотипа также может быть существлено любым способом, известным специалисту, и, в частности, путем PCR, используя соответствующие олигонуклеотиды, и секвенирования продукта PCR.
Предпочтительно был отобран клон, обладающий ожидаемыми характеристиками (orf13c:: att3Ωaac-), и назван SPM508. На деле, путем двух гибридизаций было подтверждено, что кластер att3Ωaac- действительно находится в геноме этого клона и что в самом деле достигают ожидаемого профиля рестрикции в случае замены, вследствие двойного явления рекомбинации, исходного гена копией, прерванной кластером att3Ωaac-, в геноме этого клона. Этот клон, следовательно, имеет генотип orf13c:: att3Ωaac- и был назван SPM508. Ненужным является осуществление эксцизии кластера для исследования эффекта инактивации orf13c с точки зрения ориентации генов (см. фиг.3); тот факт, что orf14 ориентирован в направлении, противоположном гену orf13c, показывает, что эти гены не являются сотранскрибированными. Использование эксцизируемого кластера, взамен, позволяет иметь возможность в любое время избавиться от маркера селекции.
Образец штамма SPM508 был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 10 июля 2002 г. под регистрационным номером I-2912.
Пример 13
Конструирование штамма Streptomyces ambofaciens, прерванного в гене orf14
Эксцизируемый кластер att3Ωaac- амплифицировали путем PCR, используя в качестве матрицы плазмиду pOSK1102 (см. выше), с помощью следующих праймеров:
EDR5: 5'GGGCGTGAAGCGGGCGAGTGTGGATGTCATGCGAGTACTCATCGCGCGCGCTTCGTTCGG 3' (SEQ ID № 100);
EDR6: 5'CGGGAAACGGCGTCGCACTCCTCGGGGGCCGCGTCAGCCCATCTGCCTCTTCGTCCCGAA 3' (SEQ ID № 101).
40 Дезоксинуклеотидов, находящихся в конце 5' этих олигонуклеотидов, включают последовательность, соответствующую последовательности в гене-мишени (в настоящем случае orf14), и 20 дезоксинуклеотидов, расположенных больше всего в 3' (представлено жирным шрифтом выше), соответствуют последовательности одного из концов эксцизируемого кластера att3Ωaac- (см. фиг.11).
Таким образом полученный продукт PCR был использован для трансформации штамма E.coli KS272, содержащего плазмиды pKOBEG и pSPM504 (см. выше), как описывается Chaveroche и др. (Chaveroche и др., 2000) (см. фиг.12 по принципу: плазмида pOS49.99 должна быть заменена плазмидой pSPM504 и полученная плазмида не является более pSPM17, а представляет собой плазмиду pSPM509). Таким образом, бактерии были трасформированы путем электропорации с помощью продукта PCR и клоны были селекционированы по их резистентности к апрамицину. Плазмиды полученных клонов были экстрагированы и рестриктированы с помощью нескольких ферментов рестрикции с целью подтверждения того, что полученный профиль рестрикции соответствует ожидаемому профилю, если имела место инсерция кластера (att3Ωaac-) в ген-мишень (orf14), то есть, если действительно произошла гомологичная рекомбинация между концами продукта PCR и гена-мишени (Chaveroche и др., 2000). Подтверждение конструкции также может быть осуществлено любым способом, известным специалисту, и, в частности, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR. Клон, плазмида которого обладает ожидаемым профилем, был отобран и соответствующая плазмида была названа pSPM509. Эта плазмида происходит от pSPM504, в которой ген orf14 прерван апрамициновым кластером (см. фиг.12). Инсерция кластера сопровождается делецией в гене orf14, прерывание начинается на уровне четвертого кодона orf14. После кластера остается последний кодон гена orf14.
Вектор pSPM509 вводили в штамм Streptomyces ambofaciens OSC2 (см. выше) путем трансформации протопластов (Kieser T. и др., 2000). После трансформации клоны селекционировали по их резистентности к апрамицину. Резистентные к апрамицину клоны затем пересевали, соответственно, на среду с апрамицином (антибиотик В) и на среду с гигромицином (антибиотик А) (см. фиг.9). Клоны, резистентные к апрамицину (ApraR) и чувствительные к гигромицину (HygS), в принципе, представляют собой такие, где имел место двойной кроссинговер и которые обладают геном orf14, прерванным кластером att3Ωaac-. Эти клоны предпочтительно были селекционированы и замена исходной копии гена orf14 копией, прерванной кластером, была подтверждена путем гибридизации. Так, полную ДНК полученных клонов рестриктировали с помощью нескольких ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом, соответствующим кластеру att3Ωaac-, для подтверждения наличия кластера в геномной ДНК полученных клонов. Вторую гибридизацию осуществляли при использовании в качестве зонда продукта PCR, соответствующего последовательности, выступающей на сотню пар оснований выше и ниже кодирующей последовательности orf14. Подтверждение генотипа также может быть существлено любым способом, известным специалисту, и, в частности, путем PCR, используя соответствующие олигонуклеотиды, и секвенирования продукта PCR.
Предпочтительно был отобран клон, обладающий ожидаемыми характеристиками (orf14:: att3Ωaac-), и назван SPM509. На деле, путем двух гибридизаций было подтверждено, что кластер att3Ωaac- действительно находится в геноме этого клона и что в самом деле достигают ожидаемого профиля рестрикции в случае замены, вследствие двойного явления рекомбинации, исходного гена копией, прерванной кластером att3Ωaac-, в геноме этого клона. Этот клон, следовательно, имеет генотип orf14:: att3Ωaac- и был назван SPM509. Ненужным является осуществление эксцизии кластера для исследования эффекта инактивации orf14 с точки зрения ориентации генов (см. фиг.3); тот факт, что orf15c ориентирован в направлении, противоположном гену orf14, показывает, что эти гены не являются сотранскрибированными. Использование эксцизируемого кластера , взамен, позволяет иметь возможность в любое время избавиться от маркера селекции.
Образец штамма SPM509 был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 10 июля 2002 г. под регистрационным номером I-2913.
Пример 14
Конструирование штамма Streptomyces ambofaciens, прерванного в гене orf6*
Инактивацию гена orf6* осуществляли с помощью способа с эксцизируемыми кластерами (см. выше и фиг.10). Для этого космиду pSPM7 использовали в качестве матрицы для амплификации фрагмента гена orf6* с помощью следующих олигонуклеотидов:
C9583: 5' CTGCAGGTGCTCCAGCGCGTCGATCT 3' (oligo sens) (SEQ ID № 102);
C9584: 5' CTGCAGACGGAGGCGGACCTGCGGCT 3' (oligo antisens) (SEQ ID № 103).
20 Дезоксинуклеотидов, находящихся в конце 3' этих олигонуклеотидов, соответствуют последовательности, расположенной в кодирующей части гена orf6* (SEQ ID № 13), и 6 дезоксинуклеотидов, находящихся более всего в 5', соответствуют последовательности сайта PstI, позволяющего впоследствии облегчать клонирование. Амплифицированный фрагмент ДНК имел размер примерно 1,11 т.н. Этот продукт PCR клонировали в векторе pGEM-T Easy (выпускается фирмой Promega (Madison, Wisconcin, США)), что позволило получить плазмиду, названную pBXL1111 (см. фиг.16).
Затем в кодирующую последовательность гена orf6* вводили эксцизируемый кластер attΩhyg+. Для этого плазмиду pBXL1111 рестриктировали с помощью ферментов рестрикции SmaI и Asp718I и продукт рестрикции обрабатывали ферментом Кленова. Эта манипуляция позволяет реализовать внутреннюю делецию 120 пар оснований в кодирующей последовательности гена orf6* (см. фиг.15). Более того, с той и другой стороны сайтов рестрикций остаются, соответственно, 511 пар оснований и 485 пар оснований последовательности orf6*, которые позволяют осуществлять гомологичную рекомбинацию для инактивации гена orf6*. Эксцизируемый кластер attΩhyg+ получали из плазмиды patt1Ωhyg+ (см. выше) путем рестрикции этой плазмиды с помощью EcoRV. Эту последнюю затем субклонировали в векторе pBXL1111, предварительно полученном как описано выше (рестрикция с помощью SmaI и Asp718I, затем обработка с помощью фермента Кленова). Полученная плазмида была названа pBXL1112 (см. фиг.17). В этой конструкции ген orf6* включает делецию из 120 пар оснований и прерван кластером attΩhyg+.
Плазмиду pBXL1112 затем рестриктировали с помощью фермента PstI (сайт, фланкирующий кластер, поскольку находится в олигонуклеотидах PCR) и вставку PstI длиной 3,7 т.н., включающую часть orf6*, прерванного кластером attΩhyg+, затем клонировали на уровне сайта PstI плазмиды pOJ260 (см. выше). Таким образом полученная плазмида была названа pBXL1113.
Вектор pBXL1113 вводили в штамм Streptomyces ambofaciens OSC2 (см. выше) путем трансформации протопластов (Kieser T. и др., 2000). После трансформации клоны селекционировали по их резистентности к гигромицину. Резистентные к гигромицину клоны затем пересевали, соответственно, на среду с гигромицином (антибиотик В) и на среду с апрамицином (антибиотик А) (см. фиг.9). Клоны, резистентные к гигромицину (HygR) и чувствительные к апрамицину (ApraS), в принципе, представляют собой такие, где имел место двойной кроссинговер и которые обладают геном orf6*, прерванным кластером att1Ωhyg+. Эти клоны предпочтительно были селекционированы и замена исходной копии гена orf6* копией, прерванной кластером, была подтверждена путем саузерн-блоттинга. Так, полную ДНК полученных клонов рестриктировали с помощью нескольких ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом, соответствующим гену hyg (получен путем PCR), для подтверждения наличия кластера в геномной ДНК полученных клонов. Вторую гибридизацию осуществляли при использовании в качестве зонда вставки PstI-PstI, содержащей ген orf6* размером примерно 1,1 т.н. и полученной из плазмиды pBXL1111 (см. выше и фиг.16). Подтверждение генотипа также может быть существлено любым способом, известным специалисту, и, в частности, путем PCR, используя соответствующие олигонуклеотиды, и секвенирования продукта PCR.
Предпочтительно был отобран клон, обладающий ожидаемыми характеристиками (orf6*:: att3Ωhyg+), и назван SPM501. На деле, путем двух гибридизаций было подтверждено, что кластер att3Ωhyg+ действительно находится в геноме этого клона и что в самом деле достигают ожидаемого профиля рестрикции в случае замены, вследствие двойного явления рекомбинации, исходного гена копией, прерванной кластером att3Ωhyg+, в геноме этого клона. Этот клон, следовательно, имеет генотип orf6*::att3Ωhyg+ и был назван SPM501.
Образец штамма SPM501 был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 10 июля 2002 г. под регистрационным номером I-2909.
Штамм SPM501 трансформировали с помощью вектора POSV508 за счет трансформации протопластов с целью провоцирования эксцизии кластера (см. фиг.14). Плазмида pOSV508 происходит от плазмиды pWHM3 (Vara J. и др., 1989) (см. фиг.13), в которую добавлены гены xis и int pSAM2 (Boccard F. и др., 1989b), находящиеся под контролем промотора ptrc (Amann E. и др., 1988) (см. фиг.14). Введение в штамм SPM501 плазмиды pOSV508, несущей гены xis и int pSAM2, позволяет осуществлять эффективную эксцизию путем сайтспецифической рекомбинации эксцизируемого кластера между сайтами attL и attR, фланкирующими этот кластер (Raynal A. и др., 1998) (фиг.10). Среди трансформантов, селекционированных по их резистентности к тиострептону, вызываемой геном tsr, который находится в pOSV508, выбирают два, ставшие чувствительными к гигромицину, ген резистентности которых находится в кластере att3Ωhyg+; следствием эксцизии, на деле, является утрата этого гена резистентности (см. фиг.10). Плазмида pOSV508 является нестабильной и после двух последовательных пассажей на среде без антибиотика выделенные клоны пересевали на среду с тиострептоном и на среду без тиострептона. Клоны, чувствительные к тиострептону, утратили pOSV508. То, что эксцизия кластера действительно имела место в рамке считывания в гене orf6*, подтверждено с помощью PCR и секвенирования продукта PCR. Прерывание начинается со 158-го кодона, 40 кодонов подверглись делеции (120 пар оснований) и после эксцизии кластера остается характерная “рубцовая” последовательность att1 из 33 пар оснований: 5' ATCGCGCGCTTCGTTCGGGACGAAGAGGTAGAT 3' (SEQ ID № 104).
Таким образом полученный и обладающий желательным генотипом (orf6*::att1) штамм был назван SPM502.
Образец штамма SPM502 был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 10 июля 2002 г. под регистрационным номером I-2910.
Пример 15
Анализ штаммов Streptomyces ambofaciens, прерванных в генах orf2, orf3, orf8, orf10, orf12, orf13c, orf14, orf6*
Для тестирования продуцирования спирамицина различными полученными штаммами разработан микробиологический тест, основанный на чувствительности штамма Micrococcus luteus (см. Gourmelon A. и др., 1998). Используемым штаммом Micrococcus luteus является штамм, происходящий от штамма DSM1790, по своей природе чувствительного к спирамицину (этот штамм, в частности, доступен из Немецкой коллекции микроорганизмов и клеточных культур ГмбХ, DSMZ) (Брауншвейг, Германия) под номером DSM1790); используемый в настоящем тесте штамм отличается от штамма DSM1790 тем, что он резистентен к конгоцидину. Этот штамм представляет собой спонтанный мутант, получаемый путем селекции на среде, содержащей возрастающие дозы конгоцидина. Такой штамм был отобран, так как Streptomyces ambofaciens продуцирует одновременно спирамицин и конгоцидин. Для цели определения продуцирования спирамицина различными получаемыми штаммами с помощью микробиологического теста, основанного на чувствительности штамма Micrococcus luteus, необходимо располагать штаммом, резистентным к конгоцидину.
Различные тестируемые штаммы Streptomyces культивировали в колбах Эрленмейера с перегородками емкостью 500 мл, содержащих 70 мл среды MP5 (Pernodet и др., 1993). Колбы Эрленмейера с перегородками инокулировали при начальной концентрации 2,5·106 спор/мл различными штаммами S. ambofaciens и культивировали при температуре 27°С при орбитальном перемешивании 250 об/мин. Отборы 2 мл суспензий осуществляли после 48, 72 и 96 часов культивирования и центрифугировали. Различные супернатанты затем замораживали при температуре -20°С. Десятикратное разведение этих супернатантов в стерильной культуральной среде использовали для теста (см. фиг.18).
Характерный штамм Micrococcus luteus, резистентный к конгоцидину, но чувствительный к спирамицину, культивировали на среде 2TY (Sambrook и др., 1989), содержащей конгоцидин в количестве 5 мкг/мл, в течение 48 часов при температуре 37°C. Определяли оптическую плотность (DO) культуры и эту культуру разводили таким образом, чтобы установить оптическую плотность, равную 4. 0,4 мл Этой предкультуры разводили в 40 мл среды DAM5 (Difco Antibiotic Medium 5, выпускаемая фирмой Difco), предварительно доведенной до температуры примерно 45°С. Эту среду затем выливали в квадратную чашку размером 12 см х 12 см и оставляли стоять для повышения температуры до комнатной.
При использовании один раз охлажденной и отвержденной среды диски из бумаги Whatman AA (см. Gourmelen A. и др., 1998) диаметром 12 мм пропитывали с помощью 70 мкл десятикратного разведения каждого супернатанта и помещали на поверхность чашки. Диски, пропитанные раствором спирамицина различных концентраций (2-4-8 мкг/мл в культуральной среде МР5), использовали в качестве эталонного ряда. Чашки выдерживали при температуре 4°С в течение 2 часов так, чтобы дать возможность осуществиться диффузии антибиотиков в агар, затем инкубировали при температуре 37°С в течение 24-48 часов.
Если диск содержит спирамицин, то он диффундирует в агар и ингибирует рост характерного штамма Micrococcus luteus. Это ингибирование вызывает появление “гало” вокруг диска, причем это гало отражает зону, где штамм Micrococcus luteus не растет. Наличие этого гало, следовательно, является указанием на присутствие спирамицина и позволяет определить, является ли штамм S. ambofaciens, соответствующий рассматриваемому диску, продуцирующим или непродуцирующим спирамицин. Сравнение с диаметрами ингибирования, достигаемыми для эталонного ряда, позволяет получить указание на количество спирамицина, продуцируемого этим штаммом. Различные штаммы, описанные в предыдущих примерах, были использованы в этом тесте для тестирования продуцирования ими спирамицина. Полученные результаты представлены в таблице 38.
Эти результаты позволяют сделать некоторое количество выводов в том, что касается функции различных генов, участвующих в биосинтезе спирамицина. Так, ген orf3 является существенным в биосинтезе спирамицина. В самом деле, инактивация в рамке считывания в этом гене приводит к штамму OS49.67 (см. пример 6), не продуцирующему больше спирамицина. Инактивация в рамке считывния позволяет отбросить гипотезу о возможном влиянии встроенного кластера на экспрессию генов, расположенных ниже orf3.
Точно также, гены orf8 и orf10 кодируют протеины, существенные в биосинтезе спирамицина, поскольку штаммы OS49.107 и OS49.50 обладают непродуцентным фенотипом. Кроме того, в этих двух последних штаммах действительно происходит инактивация соответствующего гена, который является ответственным за этот непродуцентный фенотип, так как, с точки зрения ориентации различных рамок считывания (см. фиг.3), введенная конструкция не может обладать полярным эффектом.
Изучение штаммов, обладающих эксцизируемым кластером, также позволяет сделать некоторое количество выводов в том, что касается функции прерванных генов. Штамм SPM507 имет генотип orf12::att3Ωaac-. Ненужным является осуществление эксцизии кластера для исследования эффекта инактивации orf12 с точки зрения ориентации генов (см. фиг.3). Тот факт, что orf13c ориентирован в направлении, противоположном гену orf12, показывает, что эти гены не являются сотранскрибированными. Использование эксцизируемого кластера, взамен, позволяет иметь возможность в любое время устранить маркер селекции. Фенотип штамма SPM507 не является продуцентом, следовательно, можно сделать вывод, что ген orf12 является геном, существенным в биосинтезе спирамицина в случае S. ambofaciens.
Штамм SPM508 имеет генотип orf13c::att3Ωaac-. Ненужным является осуществление эксцизии кластера для исследования эффекта инактивации orf13c с точки зрения ориентации генов (см. фиг.3). Тот факт, что orf14 ориентирован в направлении, противоположном гену orf13c, показывает, что эти гены не являются сотранскрибированными. Использование эксцизируемого кластера, взамен, позволяет иметь возможность в любое время устранить маркер селекции. Фенотип штамма SPM508 является продуцентом, следовательно, можно сделать вывод, что ген orf13c не является геном, существенным в биосинтезе спирамицина в случае S. ambofaciens.
Штамм SPM509 имеет генотип orf14::att3Ωaac-. Ненужным является осуществление эксцизии кластера для исследования эффекта инактивации orf14 с точки зрения ориентации генов (см. фиг.3). Тот факт, что orf15c ориентирован в направлении, противоположном гену orf14, показывает, что эти гены не являются сотранскрибированными. Использование эксцизируемого кластера, взамен, позволяет иметь возможность в любое время устранить маркер селекции. Фенотип штамма SPM509 не является продуцентом, следовательно, можно сделать вывод, что ген orf14 является геном, существенным в биосинтезе спирамицина в случае S. ambofaciens.
Штамм SPM21 имеет генотип orf2::att3Ωaac-. Фенотип этого штамма не является продуцентом спирамицинов. Однако ориентация генов orf1 - orf8 (см. фиг.3) позволяет полагать, что эти гены являются сотранскрибированными. Таким образом, наблюдаемый фенотип может возникать вследствие полярного воздействия кластера, встроенного в orf2, на экспрессию генов, расположенных ниже в опероне. Штамм SPM22 имеет генотип orf2::att3 и был получен после эксцизии из рамки считывания встроенного кластера. В результате эксцизии кластера остается только характерная “рубцовая” последовательность (см. пример 10). В случае штамма SPM22, также имеющего не являющийся продуцентом фенотип, можно сделать вывод, что ген orf2 представляет собой ген, существенный в биосинтезе спирамицина в S. ambofaciens. В этом случае наблюдают только эффект, вызванный инактивацией orf2.
Штамм SPM501 имеет генотип orf6*::att3Ωhyg+. Фенотип этого штамма не является продуцентом спирамицинов. Однако, так как гены orf5* и orf6* (см. фиг.3) имеют одинаковую ориентацию, наблюдаемый фенотип может возникать вследствие полярного воздействия встроенного в orf6* кластера на экспрессию orf5*. Конструкция этих генов позволяет полагать, что они могут быть сотранскрибированными. Для ответа на этот вопрос, штамм SPM502 получали после эксцизии из рамки считывания встроенного кластера. В этом штамме наблюдают только эффект инактивации orf6*. Этот штамм имеет имеет генотип orf6*::att1 (см. пример 14). В результате эксцизии кластера в рамке считывания остается только характерная “рубцовая” последовательность (см. пример 14). Штамм SPM502 обладает фенотипом-продуцентом (этот штамм, однако, продуцирует только спирамицин I (см. пример 16)). Следовательно, можно сделать вывод, что ген orf5* представляет собой ген, существенный в биосинтезе спирамицина в S. ambofaciens, так как его непрямая инактивация в штамме SPM501 приводит к фенотипу, не являющемуся продуцентом. Напротив, ген orf6* не представляет собой ген, существенный в биосинтезе спирамицина I в S. ambofaciens (напротив, он является существенным в продуцировании спирамицинов II и III (см. пример 16)).
Пример 16
Количественное определение продуцирования спирамицинов I, II и III в полученных мутантных штаммах
Различные тестируемые штаммы культивировали, каждый, в 7 колбах Эрленмейера с перегородками емкостью 500 мл, содержащих 70 мл среды MP5 (Pernodet и др., 1993). Колбы Эрленмейера инокулировали с помощью 2,5·106 спор/мл различных штаммов S. ambofaciens и культивировали при темературе 27°С при орбитальном перемешивании 250 об/мин в течение 72 часов. Культуры, соответствующие одному и тому же клону, объединяли, в случае необходимости, фильтровали через складчатый фильтр и центрифугировали в течение 15 минут со скоростью 7000 об./мин. Различные супернатанты затем хранили при температуре -30°С.
Определения осуществляли путем высокоэффективной жидкостной хроматографии (ВЭЖХ) по подбору пар ионов. Анализ путем ВЭЖХ культуральной среды позволяет точно определить концентрацию трех форм спирамицина. Используемую колонку (Macherey-Nagel) заполняли фазой Nucleosil (диоксид кремния с привитыми октильными группами). Гранулометрия составляет 5 мкм и размер пор имеет величину 100 Å. Внутренний диаметр колонки равен 4,6 мм и ее длина составляет 25 см. Подвижной фазой является буферная смесь Н3РО4 (рН=2,2) и ацетонитрила в соотношении 70:30 (по объемам), содержащая 6,25 г/л перхлората натрия (NaClO4). Анализ осуществляли в изократическом режиме с расходом, фиксированным при 1 мл/мин. Колонку терморегулировали при температуре 23°С. Детектирование обеспечивали путем УФ-спектрометрии при длине волны 238 нм. Образец охлаждали до температуры +10° и количественное определение осуществляли по площади пиков (с внутренним эталонированием). В этих условиях времена удерживания спирамицинов I, II и III составляют, соответственно, примерно 17, 21 и 30 минут, как это было подтверждено при использовании продажного образца, включающего три формы спирамицина.
Штамм OSC2 имеет продуцентный фенотип спирамицина. Это “родительский” штамм, используемый для получения штаммов, обладающих эксцизируемым кластером (см. пример 15). Этот штамм, следовательно, использовали в качестве положительного контроля продуцирования трех форм спирамицина. Этот штамм действительно продуцирует три формы спирамицинов, как было подтверждено с помощью ВЭЖХ (см. фиг.19).
Исследование штаммов, обладающих эксцизируемым кластером, позволяет сделать некоторое количество выводов в том, что касается функции прерванных генов. Штамм SPM507 имеет генотип orf2::att3Ωaac-. Фенотип штамма SPM507 не является продуцентом (см. пример 15), следовательно, можно сделать вывод, что ген orf12 является геном, существенным в биосинтезе спирамицина в случае S. ambofaciens. Этот штамм не продуцирует больше спирамицина, как подтверждено с помощью ВЭЖХ (см. фиг.22). Этот результат подтверждает существенный характер гена orf12 в биосинтезе спирамицина.
Штамм SPM508 имеет генотип orf3::att3Ωaac-. Фенотип штамма SPM508 является продуцентом спирамицина (см. пример 15), следовательно, можно сделать вывод, что ген orf13c не является геном, существенным в биосинтезе спирамицина в случае S. ambofaciens. Этот штамм продуцирует спирамицины I, II и III, как подтверждено с помощью ВЭЖХ (см. фиг.23). Этот результат подтверждает, что ген orf13c не является геном, существенным в биосинтезе спирамицинов I, II и III в случае S. ambofaciens.
Штамм SPM509 имеет генотип orf4::att3Ωaac-. Фенотип штамма SPM509 не является продуцентом, следовательно, можно сделать вывод, что ген orf14 является геном, существенным в биосинтезе спирамицина в случае S. ambofaciens. Этот штамм не продуцирует больше спирамицинов, как подтверждено с помощью ВЭЖХ (см. фиг.24). Этот результат подтверждает существенность гена orf14 в биосинтезе спирамицина.
Штамм SPM501 имеет генотип orf6*::att3Ωhyg+. Фенотип этого штамма не является продуцентом спирамицинов. Этот штамм не продуцирует больше спирамицинов, как подтверждено с помощью ВЭЖХ (см. фиг.20). Однако, так как гены orf5* и orf6* (см фиг.3) имеют одинаковую ориентацию, наблюдаемый фенотип может быть вызван за счет полярного воздействия кластера, встроенного в orf6*, на экспрессию orf5* в опероне. Это позволяет полагать, что эти гены являются сотранскрибированными. Для ответа на этот вопрос, штамм SPM502 получали путем эксцизии встроенного кластера, приводящей к делеции в рамке считывания в гене orf6* и восстанавливающей экспрессию orf5*. Этот штамм имеет генотип orf6*::att1 (см. примеры 14 и 15). В результате эксцизии кластера в рамке считывания остается только “рубцовая” последовательность att (см. пример 14). Штамм SPM502 имеет продуцентный генотип спирамицина. Однако, как обнаружено путем ВЭЖХ, этот штамм продуцирует только спирамицин I и не продуцирует спирамицинов II и III (см. фиг.21). Следовательно, из этих результатов можно сделать вывод, что ген orf5* является геном, существенным в биосинтезе спирамицина в случае S. ambofaciens, поскольку его непрямая инактивация в штамме SPM501 приводит к фенотипу, не являющемуся продуцентом спирамицина (см. фиг.20). Напротив, ген orf6* не является геном, существенным в биосинтезе спирамицина I в случае S. ambofaciens, поскольку инактивация этого гена приводит к фенотипу, являющемуся продуцентом спирамицина I (см. фиг.21). Однако orf6* яляется существенным в продуцировании спирамицинов II и III (см. пример 16). Ген orf6*, следовательно, действительно кодирует ацилтрансферазу, ответственную за модификацию платенолида в положении 3 (см. фиг.1).
Пример 17
Определение сайта инициации трансляции orf10 и повышение продуцирования спирамицинов
17.1. Конструирование плазмид pSPM523, pSPM524 и pSPM525
Ген prf10 был идентифицирован в Streptomyces ambofaciens и назван srmR согласно Geistlich и др. (Geistlich М. и др., 1992). Была осуществлена инактивация гена orf10 (см. пример 8). Так, показано, что полученный в результате штамм не продуцирует больше спирамицинов (см. пример 15). Это подтверждает, что ген orf10 действительно участвует в биосинтезе спирамицинов. Протеин, кодируемый этим геном, следовательно, в самом деле является существенным в биосинтезе спирамицинов. Однако анализ последовательности показывает, что для трансляции orf10 (см. фиг.28) могут быть использованы два кодона ATG, расположенных в одной и той же рамке считывания. Один из двух возможных кодонов (расположенный выше всего) начинается с положения 10656 последовательности, представленой в виде SEQ ID № 1, тогда как другой возможный кодон, расположенный ниже, начинается с положения 10809 (см. SEQ ID № 1). До тестирования возможного воздействия сверхпродуцирования srmR на продуцирование спирамицина важно определить, во-первых, сайт инициации трансляции.
С целью определения сайта инициации трансляции были реализованы три конструкции, включающие три формы orf10. Эти последние были получены путем PCR при использовании олигонуклеотидов, включающих либо сайт рестрикции HindIII, либо сайт рестрикции BamHI.
Первая пара праймеров, используемая для амплификации, соответствует следующим олигонуклеотидам:
EDR39 : 5'CCCAAGCTTGAGAAGGGAGCGGACATTCATGGCCCGCGCCGAACGC3' (SEQ ID № 122) (сайт HindIII выделен жирным шрифтом);
EDR42 : 5'CGGGATCCGGCTGACCATGGGAGACGGGCGCATCGCCGAGTTCAGC3' (SEQ ID № 123) (сайт BamHI выделен жирным шрифтом).
Пара праймеров EDR39-EDR42 позволяет осуществлять амплификацию фрагмента orf10, включающего ATG, расположенный больше всего в 3' (положение 10809 в последовательности, представленной в виде SEQ IDF № 1) (см. фиг.28). Полученный фрагмент имеет размер примерно 2 т.н. и впоследствии будет называться “короткий orf10”, он не содержит промотора orf10. Этот фрагмент длиной 2 т.н. клонировали в векторе pGEM-T easy для получения плазмиды pSPM520.
Вторая пара праймеров, используемая для амплификации, соответствует следующим олигонуклеотидам:
EDR40 :
5'CCCAAGCTTGAGAAGGGAGCGGACATTCAATGCTTTGGTAAAGCAC3' (SEQ ID № 124) (сайт HindIII выделен жирным шрифтом);
EDR42 : 5'CGGGATCCGGCTGACCATGGGAGACGGGCGCATCGCCGAGTTCAGC3' (SEQ ID № 123) (сайт BamHI выделен жирным шрифтом).
Пара праймеров EDR40-EDR42 позволяет осуществлять амплификацию фрагмента orf10, включающего ATG, расположенный больше всего в 5' (положение 10656 в последовательности, представленной в виде SEQ IDF № 1) (см. фиг.28). Этот фрагмент длиной 2,2 т.н., называемый впоследствии как “длинный orf10”, клонировали в векторе pGEM-T easy для получения плазмиды pSPM521.
Третья пара праймеров, используемая для амплификации, соответствует следующим олигонуклеотидам:
EDR41 :
5'-CCCAAGCTTTCAAGGAACGACGGGGTGGTCAGTCAAGT-3' (SEQ ID N° 125) (сайт HindIII выделен жирным шрифтом);
EDR42 :
5'CGGGATCCGGCTGACCATGGGAGACGGGCGCATCGCCGAGTTCAGC-3' (SEQ ID N° 123) (сайт BamHI выделен жирным шрифтом).
Пара праймеров EDR41-EDR42 позволяет осуществлять амплификацию фрагмента orf10 с двумя ATG, также как с его собственным промотором (см. фиг.28). Этот фрагмент длиной 2,8 т.н., называемый впоследствии как “orf10 pro”, клонировали в векторе pGEM-T easy для получения плазмиды pSPM522.
Фрагмент “orf10 pro” получали при использовании в качестве матрицы хромосомной ДНК штамма OSC2. Фрагменты “короткий orf10” и “длинный orf10”, в том, что касается них, получали при использовании в качестве матрицы ДНК фрагмента “orf10 pro”, предварительно очищенного.
Вставки HindIII-BamHI плазмид pSPM520, pSPM521 и pSPM522 затем субклонировали в векторе pUWL201 (плазмида, происходящая от плазмиды pUWL199 (Wehmeier U.F., 1995), в которую был встроен фрагмент KpnI-BamHI участка промотора ermE (см. Bibb и др., 1985, особенно фиг.2), обладающего мутацией, которая увеличивает “силу” промотора (промотор ermE*) (Bibb и др., 1994); (см. Doumith и др., 2000)), предварительно рестриктированном ферментами HindIII-BamHI. Таким образом были получены три плазмиды: pSPM523 (происходящая от pUWL201 с формой “короткий orf10” в качестве вставки), pSPM524 (происходящая от pUWL201 с формой “длинный orf10” в качестве вставки) и pSPM525 (происходящая от pUWL201 с формой “orf10 pro”) (фиг.28).
17.2. Трансформация штамма OS49.50 c помощью конструкций pSPM523, pSPM524 и pSPM525
Штамм OS49.50 (штамм, прерванный в гене orf10, см. пример 8) независимо трансформировали за счет трансформации протопластов (Kieser T. и др., 2000) каждой из плазмид pSPM523, pSPM524 и pSPM525. Также реализовали отрицательный контроль, трансформируя штамм OS49.50 с помощью плазмиды pUWL201 без вставки. После трансформации протопластов клоны селекционировали по их резистентности к тиострептону. Трансформация клонов с помощью каждой из плазмид была подтверждена путем экстракции этих плазмид. Таким образом были получены четыре новых штамма: штамм OSC49.50 pUWL201, происходящий от трансформации с помощью плазмиды pUWL201 без вставки; штамм OSC49.50 pSPM523, происходящий от трансформации с помощью плазмиды pSPM523; штамм OSC49.50 pSPM524, происходящий от трансформации с помощью плазмиды pSPM524; и, наконец, штамм OSC49.50 pSPM525, происходящий от трансформации с помощью плазмиды pSPM525.
Продуцирование спирамицинов каждым из этих четырех штаммов тестировали с помощью ВЭЖХ. Для этого различные тестируемые штаммы S. ambofaciens культивировали в колбах Эрленмейера с перегородками емкостью 500 мл, содержащих 70 мл среды МР5 (Pernodet и др., 1993). Когда штамм содержит плазмиду pUWL201 или одно из ее производных, добавляли 5 мкг/мл тиострептона. Колбы Эрленмейера с перегородками инокулировали при начальной концентрации 2,5·106 спор/мл различными штаммами S. ambofaciens и культуры инкубировали при температуре 27°С при орбитальном перемешивании 250 об/мин в течение 96 часов. Клетки затем отделяли от среды путем центрифугирования и супернатант анализировали путем ВЭЖХ (см. пример 16) для определения количества продуцированного спирамицина. С помощью эталонного образца и путем опроеделения площади пиков можно было определить количество каждого из спирамицинов, продуцированных этими штаммами. Результаты этого анализа представлены в таблице 39, данные выражены в мг на литр супернатанта. Результаты соответствуют общему продуцированию спирамицинов (получаемому путем суммирования продуцирования спирамицонов I, II и III).
Как показывают результаты, приведенные в таблице 39, отрицательный контроль (штамм OS49.50, трансформированный плазмидой pUWL201) не продуцирует спирамицина. Когда в штамм OS4.50 введена плазмида pSPM523 (которая содержит форму “короткий orf10”), никакого продуцирования спирамицина не наблюдают. Взамен, наличие плазмиды pSPM524 (которая содержит форму “длинный orf10”) и плазмиды pSPM525 (которая содержит форму “orf10 pro”) восстанавливает продуцирование спирамицина в штамме-хозяине OS49.50. Таким образом, единственные фрагменты orf10, содержащие полностью расположенный выше ATG, позволяют восстановить синтез спирамицина.
С целью подтверждения этих результатов, плазмиду pSPM521 (плазмида pGEM-T easy, содержащая форму “длинный orf10”) рестриктировали с помощью фермента рестрикции XhoI (этот фермент обладает единственным сайтом в этой плазмиде, локализованным между двух ATG (см. фиг.28)). Концы XhoI затем высвобождали путем обработки ферментом Кленова. Плазмиду тогда “замыкали” на самой себе путем воздействия Т4 ДНК-лигазы для получения плазмиды pSPM527. Если ATG, расположенный целиком выше (положение 10656 в последовательности, представленной в виде SEQ ID № 1), действительно используется в качестве сайта инициации трансляции, эта обработка будет вызывать разрыв рамки считывания на уровне сайта XhoI и иметь следствием продуцирование протеина, не обладающего активирующей активностью. Напротив, если инициация трансляции происходит на уровне ATG, расположенного полностью ниже (положение 10809 в последовательности, представленной в виде SEQ ID № 1), то эта обработка будет оказывать мало или вообще не будет оказывать воздействия на экспрессию Orf10 (учитывая локализацию сайта инициации транскрипции) и не будет оказывать никакого воздействия на продуцируемый протеин.
Вставку BamHI-HimdIII pSPM527 затем субклонировали в векторе pUWL201 для получения плазмиды pSPM528. Эту плазмиду встраивали в штамм OS49.50 и в особенности отбирали клон, обладающий желательной плазмидой. Продуцирование спирамицина полученным в результате штаммом затем определяли путем ВЭЖХ (см. пример 16 выше). В противоположность тому, что наблюдали в случае плазмиды pSPM524 (см. таблицу 39), наличие плазмиды pSPM528 в штамме OS49.50 не восстанавливает продуцирования спирамицина. Это подтверждает то, что началом трансляции гена orf10 является ATG, расположенный полностью ниже (ATG 1, см. фиг.28).
17.3. Повышение продуцирования спирамицинов штаммом OSC2 S. ambofaciens
Для тестирования воздействия сверхпродуцирования гена orf10 на продуцирование спирамицинов, плазмиды pSPM523, pSPM524 pSPM525 и pSPM528 вводили в штамм OSC2. Для этого протопласты штамма OSC2 трансформировали (Kieser T. и др., 2000), независимо, с помощью каждой из плазмид pSPM523, pSPM524 pSPM525 и pSPM528. Также был реализован отрицательный контроль путем трансформации штамма OSC2 c помощью плазмиды pUWL201 без вставки. После трансформации протопластов клоны селекционировали по их резистентности к тиострептону. Таким образом были получены пять новых штаммов:штамм OSC2 pUWL201, происходящий от трансформации с помощью плазмиды pUWL201 без вставки; штамм OSC2 pSPM523, происходящий от трансформации с помощью плазмиды pSPM523; штамм OSC2 pSPM524, происходящий от трансформации с помощью плазмиды pSPM524; штамм OSC2 pSPM525, происходящий от трансформации с помощью плазмиды pSPM525; и, наконец, штамм OSC2 pSPM528, происходящий от трансформации с помощью плазмиды pSPM528. Затем путем ВЭЖХ анализировали продуцирование спирамицинов этими штаммами (таким же образом, как согласно примеру 17.2). Также для сравнения параллельно осуществляли анализ продуцирования спирамицина штаммом OSC2. Результаты этого анализа представлены в таблице 40; данные выражены в мг на литр супернатанта. Результаты соответствуют общему продуцированию спирамицинов (получаемому путем суммирования продуцирования спирамицинов I, II и III).
Таким образом, наблюдали, что наличие плазмиды pSPM524 или pSPM525 значительно повышает продуцирование спирамицинов штаммом OSC2. Это на самом деле показывает, что сверхпродуцирование Orf10 оказывает положительное воздействие на продуцирование спирамицинов. Плазмида pSPM528, напротив, не оказывает воздействия на продуцирование спирамицинов.
Плазмиды pSPM525 и pUWL201 таким же образом были введены в штамм SPM502 (см. пример 14). Таким образом были получены два новых шитамма: штамм SPM502pUWL201, происходящий от трансформации с помощью плазмиды pUWL201 без вставки, и штамм PSM502pSPM525, происходящий от трансформации с помощью плазмиды pSPM525.
Образец штамма SPM502 pSPM525 (этот штамм содержит плазмиду pSPM525, см. выше) был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 26 февраля 2003 г. под регистрационным номером I-2977.
Продуцирование спирамицинов штаммами SPM502pUWL201 и SPM502pSPM525 анализировали путем ВЭЖХ (таким же образом, как согласно примеру 17.2). Также для сравнения паралельно был осуществлен анализ продуцирования спирамицинов штаммом SPM502. Результаты этих анализов представлены в таблице 42; данные выражены в мг на литр супернатанта. Результаты соответствуют продуцированию спирамицина I. В самом деле, ни один из этих штаммов не продуцирует спирамицинов II и III.
Таким образом, обнаружено, что сверхпродуцирование гена orf10 в штамме SPM502 значительно повышает продуцирование спирамицина I.
Пример 18
Конструирование банка геномных ДНК штамма OSC2 Streptomyces ambofaciens в E.coli в космиде pWED2
18.1. Конструкция космиды pWED2
С целью облегчения инактивации генов в Streptomyces конструировали космиду, несущую последовательность oriT плазмиды RK2 (что позволяет ее введение путем конъюгации в Streptomyces, исходя из соответствующего штамма E.coli) и содержащую также ген резистентности к антибиотику, придающий Streptomyces обнаружимый фенотип. Такая космида, содержащая большие вставки геномной ДНК Streptomyces ambofaciens, может быть использована в эспериментах по инактивации генов.
Для конструирования этого вектора, кластер pac-oriT (фрагмент EcoRV) встраивали в космиду pWEDl (Gourmelen и др., 1998), происходящую от космиды pWED15 (Wahl и др., 1987), на уровне единственного сайта HpaI. Кластер pac-oriT получали путем PCR. Для этого ген рас амплифицировали путем PCR, исходя из плазмиды pVF 10,4 (Vara и др., 1985; Lacalle и др., 1989), используя, в качестве первого праймера, праймер А (последовательность 5'-CCAGTAGATATC CCGCCAACCCGGAGCTGCAC-3' (SEQ ID № 126), где сайт рестрикции EcoRV подчеркнут и выделенная жирным шрифтом последовательность из 20 нуклеотидов соответствует области выше промотора гена рас) и, в качестве второго праймера, праймер В (последовательность 5'-GAAAAGATCCGT CATGGGGTCGTGCGCTCCTT-3' (SEQ ID № 127), которая включает на своем конце 5' последовательность из 12 нуклеотидов, соответствующую началу последовательности oriT (подчеркнута двойной линией), и последовательность из 20 нуклеотидов (выделенная жирным шрифтом), соответствующую концу гена рас (см. фиг.29, первая PCR).
Что касается гена oriT, то его амплифицировали путем PCR, исходя из плазмиды pPM803 (Mazodier P. и др., 1989), используя, в качестве первого праймера, праймер С (последовательность 5'-CACGACCCCATG ACGGATCTTTTCCGCTGCAT-3' (SEQ ID № 128)), который включает на своем конце 5' последовательность из 12 нуклеотидов, соответствующую последовательности ниже кодирующей последовательности гена рас (выделено жирным шрифтом), и последовательность из 20 нуклеотидов, соответствующую началу последовательности oriT, и, в качестве второго праймера, праймер D (последовательность 5'-GAGCCGGATATCATCGGTCTTGCCTTGCTCGT-3' (SEQ ID № 129)), который включает сайт рестрикции EcoRV (подчеркнутый одинарной линией) и последовательность из 20 нуклеотидов, соответствующую концу последовательности oriT (подчеркнуто двойной линией) (см. фиг.29, вторая PCR).
Продукт амплификации, полученный с праймерами А и В, и таковой, полученный с праймерами С и D, на одном из своих концов имеют общую последовательность из 24 нуклеотидов. Осуществляли третью PCR, смешивая оба продукта амплификации, полученные выше, и используя в качестве праймеров праймеры А и D (см. фиг.29, третья PCR). Это позволило получить продукт амплификации, соответствующий совокупности рас+oriT. Этот фрагмент рас-oriT затем клонировали в векторе pGEM-T Easy (выпускается фирмой Promega (Madison, Wisconsin, США)), что позволило получить плазмиду pGEM-T-pac-oriT. Вставку этой плазмиды затем клонировали в космиде pWED1. Для этого плазмиду pGEM-T-pac-oriT рестриктировали с помощью фермента EcoRV и вставку EcoRV, содержащую совокупность рас+oriT, встраивали в космиду pWED1, предварительно подвергнутую рестрикции с помощью фермента HpaI. Таким образом полученная космида была названа pWED2 (см. фиг.30).
Эта космида позволяет облегчать инактивацию генов в Streptomyces. В самом деле, она содержит последовательность oriT (что позволяет ее вводить путем конъюгации в Streptomyces, исходя из соответствующего штамма E.coli), но также содержит ген резистентности к антибиотику, придающий Streptomyces обнаружимый фенотип. Такая космида, содержащая большие вставки геномной ДНК Streptomyces ambofaciens, может быть использована в экспериментах по инактивации генов.
Так, космида, происходящая от pWED2, содержащая ген-мишень, может быть, например, введена в штамм E.coli KA272, содержащий плазмиду pKOBEG (см. Chaveroche и др., 2000), и кластер может быть введен в ген-мишень по технологии, описанной Chaveroche и др. (2000). Полученная этим способом космида (космида, в которой ген-мишень инактивирован) затем может быть введена в штамм E.coli, такой, как штамм S17.1, или любой другой штамм, позволяющий осуществлять перенос путем конъюгации плазмид, содержащих последовательность oriT, в Streptomyces.
После конъюгации между E.coli и Streptomyces, клоны Streptomyces, в которых исходная копия гена-мишени должна быть заменена прерванной копией, могут быть получены как описывается в примере 2.
Ген резистентности, экспрессированный в Streptomyces, который имеется в этой новой космиде, предствляет собой ген рас Streptomyces alboniger (Vara J. и др., 1985; Lacalle и др., 1989), который кодирует пуромицин-N-ацетилтрансферазу и который придает резистентность к пуромицину. Во время экспериментов по инактивации гена разыскивают клоны, в которых имело место двойное явление рекомбинации. Если из этих клонов удалить космиду pWED2, они, следовательно, опять станут чувствительными к пуромицину.
18.2. Конструирование банка геномных ДНК штамма OSC2 Streptomyces ambofaciens в E.coli в космиде pWED2
Геномную ДНК штамма OSC2 Streptomyces ambofaciens частично рестриктировали с помощью фермента рестрикции BamHI для получения фрагментов ДНК размером от примерно 35 т.н. до 45 т.н. Эти фрагменты затем клонировали в космиде pWED2, причем эта последняя была предварительно подвергнута рестрикции с помощью BamHI, затем обрабатывали щелочной фосфатазой. Смесь после лигирования затем инкапсидировали in vitro в частицы фагов лямбда с помощью системы “Packagene® Lambda DNA packaging system”, выпускаемой фирмой Promega (Madison, Wisconsin, США), следуя рекомендациям изготовителя. Полученные фаговые частицы использовали для инфицирования штамма SURE® E.coli, выпускаемого фирмой Stratagene (LaJolla, Калифорния, США). Селекцию клонов осуществляли на среде LB + ампициллин (50 мкг/мл), причем космида pWED2 придает резистентность к ампициллину.
Пример 19
Выделение космид нового банка, охватывающего область пути биосинтеза спирамицинов. Субклонирование и секвенирование фрагментов этих космид
19.1. Гибридизация в колониях геномного банка Streptomyces ambofaciens OSC2
Выделяли космиды нового банка Streptomyces ambofaciens OSC2 (см. пример 18), охватывающие область от orf1* до orf10* или часть или полностью область от orf1 до orf25c или большую область выше orf25c. Для этого осуществляли последовательные гибридизации в колониях E.coli, полученных согласно примеру 18, используя следующие 3 зонда (см. фиг.31):
- Первый используемый зонд соответствует фрагменту ДНК длиной примерно 0,8 т.н., амплифицированному путем PCR при использовании в качестве матрицы космиды pSPM5 и следующих праймеров:
ORF23c : 5'-ACGTGCGCGGTGAGTTCGCCGTTGC-3' (SEQ ID № 130) и
ORF25c : 5'-CTGAACGACGCCATCGCGGTGGTGC-3' (SEQ ID № 131).
Таким образом полученный продукт PCR содержит фрагмент начала orf23c, полностью orf24c и конец orf25c (см. фиг.31, зонд I).
- Второй используемый зонд соответствует фрагменту ДНК длиной примерно 0,7 т.н., амплифицированному путем PCR при использовании в качестве матрицы полной ДНК штамма S. ambofaciens АТСС23877 и следующих праймеров:
ORF1*c : 5'-GACCACCTCGAACCGTCCGGCGTCA-3' (SEQ ID № 132) и
ORF2*c : 5'-GGCCCGGTCCAGCGTGCCGAAGC-3' (SEQ ID № 133).
Таким образом полученный продукт PCR содержит фрагмент конца orf1*c и начала orf2*c (см. фиг.31, зонд II).
- Третий используемый зонд соответствует фрагменту EcoRI-BamHI длиной примерно 3 т.н., содержащий orf1, orf2 и orf3 и получаемый путем рестрикции плазмиды pOS49.99 (см. фиг.31, зонд III).
Примерно 2000 клонов банка, полученного согласно примеру 18.2, переносили на фильтр для гибридизации колоний согласно классическим способам (Sambrook и др., 1989).
Первый зонд (см. фиг.31, зонд I) метили с помощью 32Р по способу “произвольного примирования” (набор, выпускаемый фирмой Roche) и использовали для гибридизации 2000 клонов банка, после переноса на фильтр. Гибридизацию осуществляли при температуре 65°С в буфере, описанном Church и Gilbert (Church & Gilbert, 1984). Осуществляли две промывки в 2Х SSC с 0,1% додецилсульфата натрия при температуре 65°, первую - в течение 10 минут и вторую - в течение 20 минут, а третью промывку в течение 30 минут затем осуществляли в 0,2Х SSC с 0,1% додецилсульфата натрия при температуре 65°С. В этих условиях гибридизации и промывки 20 клонов из 2000 гибридизованных обладали сильным сигналом гибридизации с первым зондом. Эти 20 клонов культивировали на среде LB + ампициллин (50 мкг/мл) и 20 соответствующих космид экстрагировали путем щелочного лизиса (Sambrook и др., 1989), затем их подвергали рестрикции с помощью фермента рестрикции BamHI. Продукты рестрикций затем разделяли на геле агарозы, переносили на мембрану Nylon и гибридизировали с первым зондом (см. выше: продукт PCR ORF23c-ORF25c, зонд I) в тех же условиях, что и указанные выше. Двенадцать космид обладали фрагментом BamHI, в высокой степени гибридизирующимся с используемым зондом. Эти 12 космид были названы как pSPM34, pSPM35, pSPM36, pSPM37, pSPM38, pSPM39, pSPM40, pSPM41, pSPM42, pSPM43, pSPM44 и pSPM45. Профили, после рестрикции с помощью BamHI, этих 12 космид были сравнены между собой и профилем космиды pSPM5. Кроме того, эксперименты по амплификации путем PCR при использовании различных праймеров, соответствующих различным генам, уже идентифицированным в области orf1 - orf25c, позволили установить вставку некоторых из этих космид одних по отношению к другим и также определить локализацию этих вставок в уже известной области orf1-orf25c (см. фиг.32). В особенности была выбрана космида pSPM36, так как она способна содержать большую область выше orf25c (см. фиг.31 и 32).
Во-вторых, и используя такие же условия, как таковые, описанные выше, 2000 клонов банка Streptomyces ambofaciens OSC2 гибридизировали со вторым зондом, соответствующим продукту PCR ORF1*c-ORF2*c (см. фиг.31, зонд II). Эта гибридизация позволила выделить космиды, вставка которых расположена в области orf1*c - orf10*c. В используемых условиях гибридизации 16 клонов из 2000 гибридизированных имели сильный сигнал гибридизации с этим вторым зондом. Эти 16 клонов культивировали на среде LB+ампициллин (50 мкг/мл) и 16 соответствющих космид экстрагировали путем щелочного лизиса (Sambrook и др., 1989), затем их рестриктировали с помощью фермента рестрикции BamHI. Профили рестрикции (после рестрикции с помощью BamHI) этих 16 космид были сравнены между собой и профилем космиды pSPM7. Эксперименты по амплификации путем PCR при использовании праймеров ORF1*c и ORF2*c позволили выбрать космиды, которые действительно включали гены orf1*c и orf2*c и профили которых обладали общими зонами, однако, также и разными зонами. Кроме того, другие эксперименты по амплификации с помощью PCR при использовании различных праймеров, соответствующих различным генам, уже идентифицированным в области orf1*c - orf10*c, позволили установить вставку этих космид одних по отношению к другим и определить также локализацию этих вставок в уже известной области orf1*c - orf10*c (см. фиг.32). Отобранные в особенности две космиды были названы pSPM47 и pSPM48 (см. фиг.32).
Используя такие же условия, как таковые, описанные выше, 2000 клонов банка Streptomyces ambofaciens OSC2 также гибридизировали с третьим зондом, соответствующим фрагменту ДНК EcoRI-BamHI плазмиды pOS49.99 (см. фиг.31, зонд III). Эта гибридизация позволила выделить космиды, содержащие область от orf1 вплоть до orf3 и способные содержать либо большую часть генов PKS, либо большую часть генов orf1 - orf25c пути биосинтеза спирамицинов. В этих условиях гибридизации 35 клонов из 2000 гибридизированных имели сильный сигнал гибридизации с третьим зондом. Эти 35 клонов культивировали на среде LB+ампициллин (50 мкг/мл) и 35 соответствующих космид экстрагировали путем щелочного лизиса (Sambrook и др., 1989), затем рестриктировали с помощью фермента рестрикции BamHI. Профили, после рестрикции с помощью BamHI, этих 35 космид были сравнены между собой и профилем космиды pSPM5. Кроме того, эксперименты по амплификации путем PCR при использовании праймеров, соответствующих различным генам, уже идентифицированным в области orf1-orf25c, позволили подтвердить, что эти космиды действительно включают вставки, происходящие от области orf1-orf25c, и установить вставки одних по отношению к другим и по отношению к уже известной области orf1-orf25c (см. фиг.32). В особенности были отобраны пять космид, они были названы pSPM50, pSPM51, pSPM53, pSPM55 и pSPM56 (см. фиг.32).
19.2. Субклонирование и секвенирование одной части вставки космиды pSPM36
Зонд длиной примерно 0,8 т.н., получаемый путем PCR с праймерами ORF23c и ORF25c (см. выше и фиг.31, зонд I), также был использован в экспериментах по саузерн-блоттингу полной ДНК S. ambofaciens OSC2, рестриктированной ферментом PstI. В условиях гибридизации, описанных выше, этот зонд имеет единственный фрагмент PstI длиной примерно 6 т.н., когда гибридизуется с полной ДНК S. ambofaciens OSC2, рестриктированной с помощью фермента PstI. Существует один сайт PstI на уровне orf23c (см. SEQ ID № 80), но никакого другого сайта PstI нет вплоть до конца (сайт BamHI) известной последовательности (см. SEQ ID № 1). Этот фрагмент PstI-BamHI имеет размер примерно 1,4 т.н. Фрагмент PstI длиной 6 т.н., гибридизированный с полной ДНК S. ambofaciens, рестриктированной с помощью фермента PstI, следовательно, содержит область длиной примерно 4,6 т.н., раположенную выше orf25c. Эта область способна содержать другие гены, вырабатываемые которыми продукты участвуют в пути биосинтеза спирамицина. Путем рестрикции подтверждено, что космида pSPM36 действительно содержит этот фрагмент PstI длиной 6 т.н. Этот фрагмент был выделен из pSPM36 с целью определения последовательности, расположенной выше orf25c. Для этого космиду pSPM36 рестриктировали с помощью фермента рестрикции PstI. Фрагмент PstI-PstI длиной примерно 6 т.н. выделяли путем электроэлюции из 0,8%-ного геля агарозы, затем клонировали в векторе pBK-CMV (4512 пар оснований) (выпускается фирмой Stratagene (La Jolla, Калифорния, США)). Таким образом полученная плазмида была названа pSPM58 (см. фиг.33) и была определена последовательность ее вставки. Последовательность этой вставки представлена в виде последовательности SEQ ID № 134. Однако вся последовательность не была определена и существует разрыв длиной примерно 450 нуклеотидов, неопределенная часть последовательности была обозначена как последовательность “N” в соответствующей последовательности.
19.3. Анализ новых нуклеотидных последовательностей, определение открытых рамок считывания и характеристика генов, участвующих в биосинтезе спирамицинов
Последовательность вставки полученной космиды pSPM58 была проанализирована с помощью программы FramePlot (Ishikawa J. & Hotta K., 1999). Это позволило идентифицировать среди открытых рамок считывания открытые рамки считывания, в которых используются типичные кодоны Streptomyces. Этот анализ позволил определить, что эта вставка включает 4 новые открытые рамки считывания выше orf25c (см. фиг.34). Эти гены были названы orf26 (SEQ ID № 107), orf27 (SEQ ID № 109), orf28c (SEQ ID № 111; последовательность этой открытой рамки считывания не была определена полностью, так как существует разрыв длиной примерно 450 нуклеотидов во время секвенирования вставки pSPM58, причем эти 450 нуклеотидов фигурируют в виде ряда “N” в последовательности SEQ ID № 111) и orf29 (последовательность этой последней открытой рамки считывания является неполной в этой вставке). Буква “с”, добавленная в название гена, для рассматриваемой открытой рамки считывания означает, что кодирующая последовательность имеет обратную ориентацию (см. фиг.34).
Протеиновые последовательности, следующие из этих открытых рамок считывания, были сравнены с таковыми, находящимися в различных базах данных, с помощью различных программ: BLAST (Altschul и др., 1990; Altschul и др., 1997), CD-search (эти тдве программы доступны, в частности, из NCBI (Национальный центр информации по биотехнологии) (Bethesda, Maryland, США)), FASTA ((Pearson W. R. & Lipman D. J., 1988) и (Pearson W. R., 1990)) (эта программа доступна, в частности, из центра ресурсов INFOBIOGEN, Evry, Франция). Эти сравнения позволили сформулировать гипотезы в отношении функции вырабатываемых этими генами продуктов и идентифицировать таковые, способные участвовать в биосинтезе спирамицинов.
19.4. Субклонирование и секвенирование другой части вставки космиды pSPM36
Зонд длиной примерно 0,8 т.н., получаемый путем PCR с праймерами ORF23c и ORF25c (см. выше и фиг.31, зонд I), также был использован в экспериментах по саузерн-блоттингу полной ДНК штамма OSC2, рестриктированной ферментом StuI. В условиях гибридизации, описанных выше для этого зонда, этот зонд имеет единственный фрагмент StuI длиной примерно 10 т.н., когда гибридизуется с полной ДНК штамма OSC2, рестриктированной с помощью фермента StuI. Учитывая наличие единственного сайта StuI в orf23c (см. SEQ ID № 80) и локализацию этого сайта по отношению к сайту PstI, этот фрагмент длиной 10 т.н. включает всю совокупность вышеисследованного фрагмента PstI (вставка pSPM58) и позволяет иметь доступ к еще не изученной области длиной примерно 4 т.н. (см. фиг.33). Путем рестрикции подтверждено, что космида pSPM36 действительно содержит этот фрагмент StuI длиной 10 т.н. Этот фрагмент был выделен из космиды pSPM36 с целью определения последовательности конца orf29 и других генов, находящихся выше orf29. Для этого космиду pSPM36 рестриктировали с помощью фермента рестрикции StuI. Фрагмент StuI-StuI длиной примерно 10 т.н. выделяли путем электроэлюции из 0,8%-ного геля агарозы, затем клонировали в векторе pBK-CMV (4512 пар оснований) (выпускается фирмой Stratagene (La Jolla, Калифорния, США)). Таким образом полученная плазмида была названа pSPM72 (см. фиг.33). Эту последнюю затем рестриктировали с помощью EcoRI (сайт EcoRI во вставке pSPM58) и HindIII (сайт HindIII в сайте множественного клонирования вектора, сразу после сайта StuI конца вставки) (см. фиг.33). Таким образом полученный фрагмент ДНК EcoRI-HindIII соответствует фрагменту вставки плазмиды pSPM72 (см. фиг.33) и был клонирован в векторе pBC-SK+ (выпускается фирмой Stratagene (La Jolla, Калифорния, США)), предварительно рестриктированном с помощью EcoRI и HindIII. Таким образом полученная плазмида была названа pSPM73 и была определена последовательность ее вставки. Последовательность этой вставки представлена в виде последовательности SEQ ID № 135.
Совокупность последовательностей вставок pSPM58 и pSPM73 представлена в виде последовательности SEQ ID № 106. Эта последовательность начинается от сайта PstI на уровне orf23c (см. SEQ ID № 80) и идет вплоть до сайта StuI на уровне orf32c (см. фиг.34). Последовательность orf28c (SEQ ID № 111) не является полной (см. пример 19.3), область длиной примерно 450 нуклеотидов не секвенирована, эти 450 нуклеотидов фигурируют в виде ряда “N” в последовательности SEQ ID № 106.
19.5. Анализ новых нуклеотидных последовательностей, определение открытых рамок считывания и характеристика генов, участвующих в биосинтезе спирамицинов
Неполная последовательность вставки полученной космиды pSPM73 была проанализирована с помощью программы FramePlot (Ishikawa J. & Hotta K., 1999). Это позволило идентифицировать среди открытых рамок считывания, открытые рамки считывания, в которых используются типичные кодоны Streptomyces. Этот анализ позволил определить, что эта вставка включает 4 новые открытые рамки считывания: одну неполную и три полные (см. фиг.34). Неполная открытая рамка считывания соответствует части 3' кодирующей последовательности orf29, что позволило дополнить последовательность этого гена за счет частичной последовательности той же самой открытой рамки считывания, полученной во время секвенирования вставки плазмиды pSPM58 (см. примеры 19.2 и 19.3), совокупность этих двух последовательностей таким образом позволяет получить полную последовательность orf29. Таким образом, 4 гена были названы orf29 (SEQ ID № 113), orf30c (SEQ ID № 115), orf31 (SEQ ID № 118) и orf32c (SEQ ID № 120). Буква “с”, добавленная в название гена, для рассматриваемой открытой рамки считывания означает, что кодирующая последовательность имеет обратную ориентацию (см. фиг.34).
Протеиновые последовательности, следующие из этих открытых рамок считывания (SEQ ID № 114 для orf29, SEQ ID № 116 и 117 для orf30c, SEQ ID № 119 для orf31 и SEQ ID № 121 для orf32c), были сравнены с таковыми, находящимися в различных базах данных, с помощью различных программ: BLAST (Altschul и др., 1990; Altschul и др., 1997), CD-search (эти две программы доступны, в частности, из NCBI (Национальный центр информации по биотехнологии) (Bethesda, Maryland, США)), FASTA ((Pearson W. R. & Lipman D. J., 1988) и (Pearson W. R., 1990)) (эта программа доступна, в частности, из центра ресурсов INFOBIOGEN, Evry, Франция). Эти сравнения позволили сформулировать гипотезы в отношении функции вырабатываемых этими генами продуктов и идентифицировать таковые, способные участвовать в биосинтезе спирамицинов.
19.6. Субклонирование и секвенирование третьей части вставки космиды pSPM36
Зонд (фрагмент ДНК длиной 0,8 т.н.), соответствующий внутренней последовательности orf32c, получали путем PCR при использовании в качестве матрицы полной ДНК штамма Streptomyces ambofaciens и следующих праймеров:
KF36: 5'- TTGCCGTAGCCGAGGACCAGCG-3' (SEQ ID № 151) и
KF37: 5'- CACATGGCCCTGGAGGACCCTG-3' (SEQ ID № 152).
Таким образом полученный продукт PCR имеет внутреннюю последовательность orf32c. Этот зонд был использован в экспериментах по саузерн-блоттингу хромосомной ДНК штамма OSC2 и ДНК космиды pSPM36, рестриктированных с помощью фермента PstI. Используя такие же условия гибридизации, как таковые, описанные выше (см. пример 19.1), этот зонд имеет два фрагмента PstI длиной примерно 3,4 т.н. и 2,5 т.н., когда гибридизуется с полной ДНК штамма OSC2 и ДНК космиды pSPM36, рестриктированными с помощью фермента PstI. Учитывая наличие сайта PstI в используемом зонде, можно объяснить эти результаты. Первый фрагмент ДНК, который имеет длину примерно 3,4 т.н., представляет собой фрагмент, последовательность которого уже полностью известна. Последовательность фрагмента длиной 2,5 т.н. известна только частично, в области 700 пар оснований. Этот фрагмент был выделен из космиды pSPM36 с целью определения последовательности конца orf32c и других генов выше этого последнего. Для этого космиду pSPM36 рестриктировали с помощью фермента рестрикции PstI. Фрагмент PstI-PstI длиной примерно 2,5 т.н. выделяли путем очистки из 0,8%-ного геля агарозы, затем клонировали в векторе pBK-CMV (4518 пар оснований) (выпускается фирмой Stratagene (La Jolla, Калифорния, США)). Таким образом полученная плазмида была названа pSPM79 (см. фиг.41) и была определена последовательность ее вставки.
Последовательность orf28c (SEQ ID № 111) является неполной (см. пример 19.3). В самом деле, область длиной примерно 450 нуклеотидов не была определена, эти 450 нуклеотидов фигурируют в виде ряда “N” в последовательности SEQ ID № 106. Последовательность всей в целом недостающей области была определена путем повторного секвенирования этой области. Последовательность вставок pSPM58 и pSPM73, следовательно, была определена полностью. Полная последовательность кодирующей части orf28c представлена в виде последовательности SEQ ID № 141 и протеин, следующий из этой последовательности, в виде последовательности SEQ ID № 142. Последовательность вставки плазмиды pSPM79 представлена в виде последовательности SEQ ID № 161.
Совокупность последовательностей вставок pSPM58, pSPM73 и pSPM79 представлена в виде последовательности SEQ ID № 140 (см. фиг.41). Эта последовательность начинается от сайта PstI на уровне orf23c (см. SEQ ID № 80) и идет вплоть до сайта PstI на уровне orf34c (см. фиг.41).
19.7. Анализ новых нуклеотидных последовательностей, определение открытых рамок считывания и характеристика генов, которые могут участвовать в биосинтезе спирамицинов
Последовательность вставки полученной плазмиды pSPM79 была проанализирована с помощью программы FramePlot (Ishikawa J. & Hotta K., 1999). Это позволило идентифицировать среди открытых рамок считывания открытые рамки считывания, в которых используются типичные кодоны Streptomyces. Этот анализ позволил определить, что эта вставка включает 3 открытые рамки считывания: две неполных (orf32c и orf34c) и одну полную (orf33) (см. фиг.41).
Первая неполная открытая рамка считывания соответствует части 5' кодирующей последовательности orf32c. Это позволило дополнить последовательность этого гена за счет частичной последовательности той же самой открытой рамки считывания, полученной во время секвенирования вставки плазмиды pSPM73 (см. примеры 19.4 и 19.5), совокупность этих двух последовательностей таким образом позволяет получить полную последовательность orf32c (SEQ ID № 145). Буква “с”, добавленная в название гена, для рассматриваемой открытой рамки считывания показывает, что кодирующая последовательность находится в обратной ориентации (см. фиг.41). Полная открытая рамка считывания (ORF) была названа orf33 (SEQ ID № 147). Третья ORF была названа orf34c (SEQ ID № 149). Буква “с”, добавленная в название гена, для рассматриваемой открытой рамки считывания показывает, что кодирующая последовательность находится в обратной ориентации (см. фиг.37). Сравнения, осуществленные между продуктом этой ORF и банками данных, позволяют полагать, что С-концевая часть этого протеина отсутствует в продукте, следующем из нуклеотидной последовательности, и, следовательно, что эта ORF является более длинной и продолжается вне секвенированной области.
Протеиновые последовательности, следующие из этих открытых рамок считывания, были сравнены с таковыми, находящимися в различных базах данных, с помощью различных программ: BLAST (Altschul и др., 1990; Altschul и др., 1997), CD-search (эти две программы доступны, в частности, из NCBI (Национальный центр информации по биотехнологии) (Bethesda, Maryland, США)), FASTA ((Pearson W. R. & Lipman D. J., 1988) и (Pearson W. R., 1990)) (эта программа доступна, в частности, из центра ресурсов INFOBIOGEN, Evry, Франция). Эти сравнения позволили сформулировать гипотезы в отношении функции вырабатываемых этими генами продуктов и идентифицировать таковые, способные участвовать в биосинтезе спирамицинов.
Пример 20
Анализ продуцирования медиаторов биосинтеза спирамицинов
20.1. Приготовление образцов
Различные тестируемые штаммы культивировали, каждый, в 7 колбах Эрленмейера с перегородками емкостью 500 мл, содержащих 70 мл среды MP5 (Pernodet и др., 1993). Колбы Эрленмейера инокулировали с помощью 2,5·106 спор/мл различных штаммов S. ambofaciens и культивировали при темературе 27°С при орбитальном перемешивании 250 об/мин в течение 96 часов. Культуры, соответствующие одному и тому же клону, объединяли, в случае необходимости, фильтровали через складчатый фильтр и центрифугировали в течение 15 минут со скоростью 7000 об./мин.
Значение рН супернатанта затем доводили до 9 с помощью раствора гидроксида натрия и супернатант экстрагировали с помощью метилизобутирилкетона (MIBK). Органическую фазу (MIBK) тогда рекуперировали и выпаривали. Сухой экстракт затем обрабатывали с помощью 1 мл ацетонитрила, после чего разводили в соотношении 1:10 (100 мкл до 1 мл с помощью воды) перед использованием для анализов путем жидкостной хроматографии в сочетании с масс-спектрометрией (CL/SM).
20.2. Анализ образцов путем CL/SM
Образцы анализировали путем CL/SM с целью определения массы различных продуктов, синтезированных тестируемыми штаммами.
Используемой колонкой для высокоэффективной жидкостной хроматографии (ВЭЖХ) является колонка Kromasil С8 150* с внутренним диаметром 4,6 мм; гранулометрия составляет 5 мкм и размер пор равен 100 Е.
Подвижной фазой является градиент, образованный смесью ацетонитрила и 0,05%-ного водного раствора трифторуксусной кислоты, расход фиксирован при 1 мл/мин. Температуру колонки поддерживали при 30°С.
УФ-детектирование на выходе из колонки осуществляли при двух различных длинах волн: 238 нм и 280 нм.
Масс-спектрометр, соединенный с хроматографической колонкой, представляет собой прибор Simple Quadripole, выпускаемый фирмой Agilent, с конусными напряжениями 30 вольт и 70 вольт.
20.3. Анализ медиаторов биосинтеза, продуцируемых штаммом OS49.67
Штамм OS49.67, в котором ген orf3 инактивирован путем делеции в рамке считывания, не продуцирует спирамицинов (см. примеры 6 и 15).
Образец приготовляли по способу, описанному выше (см. параграф 20.1) и анализировали с помощью CL/SM как описано выше (см. параграф 20.2).
Более конкретно, анализ путем хроматографии осуществляли с использованием градиента растворителя в качестве подвижной фазы: 20% ацетонитрила во время Т=от 0 до 5 минут, затем линейное повышение до 30% вплоть до Т=35 минут с последующим периодом устойчивости вплоть до Т=50 минут.
В этих условиях в особенности наблюдали два продукта: поглощающий при 238 нм (время удерживания 33,4 минуты) и поглощающий при 280 нм (время удерживания 44,8 минут) (см. фиг.35). На фиг.35 представлена суперпозиция хроматограмм ВЭЖХ, полученных при 238 нм и 280 нм (вверху), а также УФ-спектров молекул, элюированных при 33,4 минутах и 44,8 минутах (внизу).
Условия анализов с помощью связанной с ВЭЖХ масс-спектрометрии были следующими: поиск осуществляли методом сканирования, охватывающим гамму масс, включающую от 100 Да до 1000 Да. Увеличение электроумножителя составляло 1 вольт. Что касается источника электронного распыления, давление распыляемого газа составляло 35 фунтов/дюйм2, расход осушенного газа составлял 12,0 л/мин-1, температура осушенного газа составляла 350°С, капиллярное напряжение было доведено до ± 3000 вольт. Эти эксперименты позволили определить массу двух разделенных продуктов. Эти массы составляют, соответственно, 370 г/моль для продукта, элюированного первым ([M-H2O]+=353, основной продукт) и 368 г/моль для второго продукта ([M-H2O]+=351, основной продукт).
Для определения структуры вышеуказанные продукты были выделены и очищены в следующих условиях: подвижной фазой является смесь 0,05%-ного водного раствора трифторуксусной кислоты и ацетонитрила в соотношении 70:30 (об/об). Хроматографию осуществляли в изократическом режиме с дебитом, фиксированным при 1 мл/мин. В этих условиях времена удерживания 2 продуктов составляют, соответственно, 8 минут и 13,3 минуты. Кроме того, в этом случае, приготовленнный образец (см. параграф 20.1) не был разведен в воде перед инжекцией 10 мкл.
2 Продукта рекуперировали на выходе из хроматографической колонки и выделяли в следующих условиях: патрон Oasis HLB, 1 см3, 30 мг (Waters), кондиционировали последовательно с помощью 1 мл ацетонитрила, затем 1 мл смеси воды и ацетонитрила (20 об./80 об.) и 1 мл смеси воды и ацетонитрила в соотношении 80:20. Затем вводили образец и патрон последовательно промывали с помощью 1 мл смеси воды и ацетонитрила (95:5), 1 мл смеси воды и дейтерированного ацетонитрила (95:5), затем элюировали с помощью 600 мкл смеси воды и дейтерированного ацетонитрила в соотношении 40:60. Рекуперированный раствор затем прямо анализировали с помощью ЯМР.
Спектры ЯМР, полученные для этих двух соединений, являются следующими:
- первый элюированный продукт: платенолид А (спектр 9646V)
Спектр 1Н в CD3CN (химические сдвиги в м.д.): 0,90 (3H, т, J=6 Гц), 0,93 (3H, д, J=5 Гц), 1,27 (3H, д, J=5 Гц), между 1,27 и 1,40 (3H, м), 1,51 (1H, м), 1,95 (1H, м), 2,12 (1H, м), 2,30 (1H, д, J=12 Гц), 2,50 (1H, д, J=11 Гц), 2,58 (1H, дд, J=9 и 12 Гц), 2,96 (1H, д, J=7 Гц), 3,43 (3H, с), 3,70 (1H, д, J=9 Гц), 3,86 (1H, д, J=7 Гц), 4,10 (1H, м), 5,08 (1H, м), 5,58 (1H, дт, J=3 и 12 Гц), 5,70 (1H, дд, J=8 и 12 Гц), 6,05 (1H, дд, J=9 и 12 Гц), 6,24 (1H, дд, J=9 и 12 Гц).
- второй элюированный продукт: платенолид В (спектр 9647V)
Спектр 1Н в CD3CN (химические сдвиги в м.д.): 0,81 (3H, т, J=6 Гц), 0,89 (1H, м), 1,17 (3H, д, J=5 Гц), 1,30 (4H, м), 1,47 (2H, м), 1,61 (1H, т, J=10 Гц), 2,20 (1H, м), 2,38 (1H, д, J=13 Гц), 2,52 (1H, м), 2,58 (1H, м), 2,68 (1H, дд, J=8 и 13 Гц), 3,10 (1H, д, J=7 Гц), 3,50 (3H, с), 3,61 (1H, д, J=8 Гц), 3,82 (1H, д, J=7 Гц), 5,09 (1H, м), 6,20 (1H, м), 6,25 (1H, дд, J=9 и 12 Гц), 6,58 (1H, д, J=12 Гц), 7,19 (1H, дд, J=9 и 12 Гц).
Эти эксперименты, таким образом, позволили определить структуру этих двух соединений. Первым элюированным продуктом является платенолид А и вторым - платенолид В; установленная структура этих двух молекул представлена на фиг.36.
Также с помощью метода CL/SM, ассоциированого с ЯМР, в условиях, слегка отличных от таковых, описанных выше, но установление которых хорошо известно специалисту, определено, что штамм OS49.67 продуцирует, кроме платенолидов А и В, производное этих двух соединений. Речь идет о соединениях платенолид А + микароза и платенолид В + микароза (структура этих двух соединений представлена на фиг.40). Результаты анализа супернатанта штамма OS49/67 представлены в таблице 42.
Точная масса=370
Молекулярная формула=C20H34O6
393,0
[M+K]+
408,9
[M-H2O+H]+
353,0
[2M+Na]+
763,2
Точная масса=368
Молекулярная формула=C20H32O6
351,0
[M+Na]+
391,0
[M+K]+
406,9
[2M+Na]+
759,1
Точная масса=514
Молекулярная формула=C27H46O9
537,0
[M+K]+
553,0
Точная масса=512
Молекулярная формула=C27H44O9
535,0
[M+K]+550,9
[PlatB-H2O+H]+350,9
20.4. Анализ медиаторов биосинтеза, продуцируемых штаммом SPM509
Штамм SPM509, в котором ген orf14 инактивирован (orf14::attΩaac-), не продуцирует спирамицинов (см. примеры 13, 15 и 16). Образец был получен по способу, описанному выше (см. параграф 20.1) и проанализирован путем CL/SM, как описано выше (см. параграфы 20.2 и 20.3). Анализ медиаторов биосинтеза, присутствующих в супернатанте культуры штамма SPM509, культивированного на среде МР5, показал, что этот штамм продуцирует только форму В платенолида (“платенолид В”, см. фиг.36), но не форму А (“платенолид А”, см. фиг.36).
Пример 21
Прерывание гена orf14 в штамме, прерванном в гене orf3 (OS49.67)
Вырабатываемый геном orf14 продукт необходим в биосинтезе спирамицинов (см. примеры 13, 15 и 16: штамм SPM509, в котором этот ген прерван, не продуцирует больше спирамицинов). Анализ медиаторов биосинтеза, присутствующих в супернатанте культуры штамма SPM509, культивированного на среде МР5, показал, что этот штамм продуцирует только форму В платенолида, но не форму А (см. пример 20). Одной из гипотез, которая может объяснить это наблюдение, является то, что продукт orf14 участвует в конверсии формы В платенолида в форму А путем ферментативной стадии восстановления. Для проверки этой гипотезы ген orf14 был инактивирован с образованием мутанта, не продуцирующего больше спирамицинов, но продуцирующего формы А и В платенолида. Это случай штамма OS49.67 (см. примеры 6 и 20), в котором ген orf3 инактивирован путем делеции рамки считывания (Δorf3). Для инактивации гена orf14 в этот штамм была введена плазмида pSPM509 путем трансформации протопластов штамма OS49.67 (Rieser T. и др., 2000). Инактивация гена orf14 уже была описана в случае штамма OSC2 (см. пример 13) и таким же образом она была осуществлена для инактивации гена orf14 в штамме OS49.67. После трансформации с помощью плазмиды pSPM509 клоны селекционировали по их резистентности к апрамицину. Резистентные к апрамицину клоны затем пересевали, соответственно, на среду с апрамицином и на среду с гигромицином. Клоны, резистентные к апрамицину (ApraR) и чувствительные к гигромицину (HygS), представляют собой, в принципе, такие, где имел место двойной кроссинговер и в которых ген orf14 заменен копией orf14, прерванной кластером att3Ωaac-. В особенности эти клоны были отобраны и замена исходной копии orf14 копией, прерванной кластером, была подтверждена путем гибридизации. Так, полную ДНК полученных клонов рестриктировали с помощью нескольких ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом, соответствующим кластеру att3Ωaac-, для подтверждения того, что замена гена действительно имела место. Подтверждение генотипа также может быть реализовано любым способом, известным специалисту, и, в частности, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR.
В особенности был отобран клон, обладающий ожидаемыми характеристиками (Δorf3, orf14:: att3Ωaac-), и назван SPM510. В самом деле, благодаря двум гибридизациям было подтверждено, что кластер att3Ωaac- действительно находится в геноме этого клона и что действительно получают ожидаемый профиль рестрикции в случае замены, за счет двойной рекомбинации, исходного гена orf14 копией, прерванной кластером att3Ωaac-, в геноме этого клона.
Пример 22
Функциональная комплементация прерывания гена orf14
22.1. Конструкция плазмиды pSPM519
Ген orf14 амплифицировали путем PCR при использовании пары следующих олигонуклеотидов: EDR31 : 5' CCCAAGCTTCTGCGCCCGCGGGCGTGAA 3' (SEQ ID № 136) и EDR37 : 5' GCTCTAGAACCGTGTAGCCGCGCCCCGG 3' (SEQ ID № 137) и, в качестве матрицы, хромосомной ДНК штамма OSC2. Олигонуклеотиды EDR31 и EDR37 содержат, соответственно, сайт рестрикции HindIII и XbaI (последовательность, выделенная жирным шрифтом). Таким образом полученный фрагмент длиной 1,2 т.н. клонировали в векторе pGEM-T easy (выпускается фирмой Promega (Madison, Wisconcin, США)) для получения плазмиды pSPN515. Эту плазмиду затем рестриктировали с помощью ферментов рестрикции HindIII и XbaI. Полученную вставку HindIII/XbaI длиной 1,2 т.н. клонировали в векторе pUWL201 (см. пример 17.1), предварительном рестриктированном с помощью тех же ферментов. Таким образом полученная плазмида была названа pSPM519.
22.1. Трансформация штаммов SPM509 и SPM510 с помощью плазмиды pSPM519
Плазмиду pSPM519 вводили в штаммы SPM509 (см. пример 13) и SPM510 (см. пример 17) путем трансформации протопластов (Kieser T. и др., 2000). После трансформации клоны селекционировали по их резистентности к тиострептону. Клоны затем пересевали на содержащую тиострептон среду.
Штамм SPM509 представляет собой штамм, не являющийся продуцентом спирамицинов (см. примеры 13, 15 и 16 и фиг.24). Продуцирование спирамицинов штаммом SPM509, трансформированным вектором pSPM519 (штамм, называемый SPM509pSPM519), было проанализировано путем культивирования этого штамма на среде МР5 в присутствии тиострептона. Культуральные супернатанты затем анализировали путем ВЭЖХ (см. примеры 16 и 17). Результаты этого анализа представлены в таблице 43, данные выражены в мг на литр супернатанта. Результаты соответствуют общему продуцированию спирамицинов (получают путем суммирования продуцирования спирамицинов I, II и III). Обнаружено, что присутствие вектора pSPM519 в штамме SPM509 восстанавливает продуцирование спирамицинов (см. таблицу 43).
Штамм SPM510, трансформированный с помощью плазмиды pSPM519, был назван SPM510pSPM519.
Пример 23
Функциональная комплементация прерывания гена orf3 геном tylB S. fradiae
23.1. Конструкция плазмиды pOS49.52
Плазмида pOS49.52 соответствует плазмиде, позволяющей осуществлять экспрессию протеина TylB в S. ambofaciens. Для ее конструирования, кодирующую последовательность гена tylB S. fradiae (Merson-Davies & Cundliffe, 1994; номер доступа в банк генов: U08223 (последoвательность области), SFU08223 (последовательность ДНК) и ААА21342 (протеиновая последовательность)) встраивали в плазмиду рКС1218 (Bierman и др., 1992; Kieser и др., 2000; штамм E. coli, содержащий эту плазмиду, доступен из ARS (NRRL) (Коллекция культур службы исследований в сельском хозяйстве) (Peoria, Иллинойс, США), под номером В-14790). Кроме того, эта кодирующая последовательность находилась под контролем промотора ermE* (см. выше, в частности, пример 17.1; Bibb и др., 1985; Bibb и др., 1994).
23.1. Трансформация штамма OS49.67 с помощью плазмиды pOS49.52
Штамм OS49.67, в котором ген orf3 инактивирован путем делеции в рамке считывания, не продуцирует спирамицинов (см. примеры 6 и 15). Плазмиду pOS49.52 вводили в штамм OS49.67 путем трансформации протопластов (Kieser T. и др., 2000). После трансформации клоны селекционировали по их резистентности к апрамицину. Клоны затем пересевали на среду, содержащую апрамицин. В особенности был отобран один клон и назван OS49.67 pOS49.52.
Как было показано выше, штамм OS49.67 не продуцирует спирамицины (см. примеры 6 и 15). Продуцировние спирамицинов штаммом OS49.67, трансформированным вектором pOS49.52, было проанализировано способом, описанным в примере 15. Таким образом, было показано, что этот штамм имеет фенотип, являющийся продуцентом спирамицинов. Таким образом, протеин TylB позволяет осуществлять функциональную комплементацию прерывания гена orf3.
Пример 24
Повышение продуцирования спирамицинов путем сверхпродуцирования гена orf28c
24.1. Конструкция плазмиды pSPM75
Ген orf28c амплифицировали путем PCR, используя пару нуклеотидов, включающих сайт рестрикции HindIII или сайт рестрикции BamHI. Эти праймеры имеют следующие последовательности:
KF30: 5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID № 138) с сайтом рестрикции HindIII (который выделен жирным шрифтом);
KF31: 5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID № 139) с сайтом рестрикции BamHI (который выделен жирным шрифтом).
Праймеры KF30 и KF31 содержат, соответственно, сайт рестрикции HindIII и BamHI (последовательности, выделенные жирным шрифтом). Пара праймеров KF30 и KF31 позволяет амплифицировать фрагмент ДНК размером примерно 1,5 т.н., содержащий ген orf28c, используя в качестве матрицы космиду pSPM36 (см. выше). Таким образом полученный фрагмент длиной 1,5 т.н. клонировали в векторе pGEM-T easy (выпускается фирмой Promega (Madison, Wisconcin, США)) для получения плазмиды pSPM74. Плазмиду pSPM74 затем рестриктировали ферментами рестрикции HindIII и BamHI и полученную вставку HindIII/BamHI длиной примерно 1,5 т.н. субклонировали в векторе pUWL201 (см. пример 17.1), предварительно рестриктированном с помощью тех же ферментов. Таким образом полученная плазмида была названа pSPM75, она содержит совокупность кодирующей последовательности orf28c, находящейся под контролем промотора ermE*.
24.2. Трансформация штамма OSC2 с помощью плазмиды pSPM75
Плазмиду pSPM75 вводили в штаммы OSC2 путем трансформации протопластов (Kieser T. и др., 2000). После трансформации протопластов клоны селекционировали по их резистентности к тиострептону. Клоны затем пересевали на среду, содержащую тиострептон, и трансформацию с помощью плазмиды подтверждали путем экстракции плазмид. В особенности были отобраны два клона и названы OSC2/pSPM75(1) и OSC2/pSPM75(2).
Образец штамма OSC2/pSPM75(2) был депонирован в Национальной коллекции культур микроорганизмов (CNCM), Институт Пастера, 25, rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 6 октября 2003 г. под регистрационным номером I-3101.
Для проверки воздействия сверхпродуцирования гена orf289c на продуцирование спирамицинов продуцирование спирамицинов клонами OSC2/pSPM75(1) и OSC2/pSPM75(2) тестировали по способу, описанному в примере 15. Анализ продуцирования спирамицинов штаммом OSC2 также осуществляли параллельно для сравнения. Таким образом, было обнаружено, что наличие плазмиды pSPM75 значительно повышает продуцирование спирамицинов штаммом OSC2. Это показывает, что сверхпродуцирование orf28c приводит к повышению продуцирования спирамицинов и подтверждает его роль в качестве регулятора.
Продуцирование спирамицинов клонами OSC2/pSPM75(1) и OSC2/pSPM75(2) также было проанализировано путем ВЭЖХ (таким же образом, как указано в примере 17.2). Анализ в отношении продуцирования спирамицинов штаммом OSC2 также осуществляли параллельно для сравнения. Результаты этого анализа представлены в таблице 44, данные выражены в мг на литр супернатанта. Результаты соответствуют общему продуцированию спирамицинов (получают путем суммирования продуцирования спирамицинов I, II и III).
Таким образом, обнаружено, что присутствие плазмиды pSPM75 значительно повышает продуцирование спирамицинов штаммом OSC2. Это в самом деле показывает, что сверхпродуцирование orf28c оказывает положительное действие на продуцирование спирамицинов.
Пример 25
Анализ продуцирования медиаторов биосинтеза спирамицинов штаммом, инактивированным в гене orf8
Штамм OS49.107, в котором ген orf8 инактивирован путем инсерции кластера Ωhyg, не продуцирует спирамицинов (см. примеры 7 и 15). Ген orf8 кодирует протеин, обладающий относительно большим сходством с несколькими аминотрансферазами, и в высшей степени позволяет полагать, что ген orf8 кодирует 4-амино-трансферазу, ответственную за реакцию трансаминирования, необходимую в биосинтезе форозамина (см. фиг.6). Следовательно, ожидается, что биосинтез спирамицинов блокируется на стадии фороцидина (см. фиг.7). Штамм OS49.107, который не является продуцентом спирамицина, следовательно, должен будет продуцировать фороцидин.
Образец супернатанта штамма OS49.107 приготовляли по способу, описанному выше (см. пример 16, без экстракции с помощью MIBK), и анализировали путем CL/SM, как описано выше (см. параграфы 20.2 и 20.3). Согласно методу SIM была отобрана масса 558, относящаяся к молекулярному иону фороцидина, и было детектировано несколько пиков. Присутствие соединения массой 558 согласуется с гипотезой о роли orf8 в синтезе форозамина.
Пример 26
Анализ продуцирования медиаторов биосинтеза спирамицинов штаммом, инактивированным в гене orf12
Штамм SPM507, в котором ген orf12 инактивирован, не продуцирует спирамицинов (см. примеры 11 и 15). Ген orf12 кодирует 3,4-дегидратазу, ответственную за реакцию дегидратации, нобходимую в биосинтезе форозамина (см. фиг.6). Следовательно, ожидается, что биосинтез спирамицинов блокируется на стадии фороцидина (см. фиг.7). Штамм SPM507, который не является продуцентом спирамицина, следовательно, должен будет продуцировать фороцидин.
Образец супернатанта штамма SPM507 приготовляли по способу, описанному выше (см. пример 16, без экстракции с помощью MIBK), и анализировали путем CL/SM, как описано выше (см. параграфы 20.2 и 20.3). В этих условиях, время удерживания фороцидина составляет примерно 12,9 минут. Согласно методу SIM, была отобрана масса 558, относящаяся к молекулярному иону [M+H]+ фороцидина, и был детектирован один пик. Присутствие соединения массой 558 позволяет признать действительной гипотезу о роли вырабатываемого геном orf12 продукта в биосинтезе спирамицинов.
Однако фороцидин находится в относительно небольшом количестве, и в этих условиях в особенности был обнаружен продукт, поглощающий при 238 нм (время удерживания 17,1 мин). Анализ путем LC/SM позволил определить массу этого соединения, которая составляет 744,3 г/моль ([M+H]+=744,3; основной продукт).
Для определения структуры вышеуказанные продукты выделяли и очищали в условиях, описанных выше (см. параграф 20.1). Органическую фазу (MIBK) затем рекуперировали и выпаривали. Сухой экстракт обрабатывали водой и экстрагировали гептаном. Водный раствор затем эктрагировали путем фиксации на патроне Oasis HLB, 1 г (Waters SAS, St-Quentin en-Yvelines, Франция). Соединение рекуперировали путем элюирования смесью вода/ацетонитрил в соотношении 30:70. Этот раствор затем инжектировали (100 мкл) в аналитическую колонку и фракции собирали в патроне Oasis HLB, 1 см3, 30 мг (Waters). Перед использованием патроны Oasis HLB, 1 см3, 30 мг (Waters), кондиционировали последовательно ацетонитрилом, затем смесью вода/ацетонитрил (20 об./80 об.) и смесью вода/ацетонитрил в соотношении 80:20.
Патрон Oasis HLB, 1 см3, 30 мг (Waters), затем промывали последовательно с помощью 1 мл смеси вода/ацетонитрил (95:5), 1 мл смеси вода/дейтерированный ацетонитрил (95:5), затем элюировали с помощью 600 мкл смеси вода/дейтерированный ацетонитрил (40:60). Рекуперированный раствор затем прямо анализировали путем ЯМР.
Спектр ЯМР, полученный для этого соединения, явлется следующим (спектр ЯМР 19312V):
Спектр 1Н в CD3CN/D2O (химические сдвиги в м.д.): 0,92 (3H, д, J=6 Гц), 1,10 (1H, м), 1,14 (3H, с), 1,17 (3H, д, J=6 Гц), 1,22 (3H, д, J=6 Гц), 1,25 (3H, д, J=6 Гц), 1,40 (1H, м), 1,75 (1H, дд, J=12 и 2 Гц), 1,81 (1H, м), 1,90 (1H, д, J=12 Гц), 2,05 (1H, м), 2,12 (3H, с), 2,15 (1H, м), 2,35 (2H, м), 2,45 (6H, с больш.), 2,53 (1H, м), 2,64 (1H, дд, J=12 и 9 Гц), 2,80 (1H, дд, J=9 и 16 Гц), 2,95 (1H, д, J=8 Гц), 3,23 (2H, м), 3,34 (1H, д, J=7 Гц), 3,45 (3H, с), 3,49 (1H, м), 3,93 (1H, дд, J=7 и 3 Гц), 4,08 (1H, м), 4,37 (1H, д, J=6 Гц), 4,88 (1H, м), 5,05 (2H, м), 5,65 (2H, м), 6,08 (1H, дд, J=8 и 12 Гц), 6,40 (1H, дд, J=12 и 9 Гц), 9,60(1H, с).
Эти эксперименты, таким образом, позволили определить структуру этого соединения, она представлена на фиг.38.
Пример 27
Анализ продуцирования медиаторов биосинтеза спирамицинов штаммом, инактивированным в гене orf5*
Штамм SPM501 имеет генотип orf6*::attΩhyg+. Благодаря полярному эффекту инсерции кластера attΩhyg+ в ген orf6* определено, что ген orf5* необходим в пути биосинтеза спирамицинов. На самом деле, инсерция эксцизируемого кластера в кодирующую часть гена orf6* вызывает полное прекращение продуцирования спирамицинов за счет полярного воздействия на экспрессию гена orf5*. Однако, как только встроенный кластер был эксцизирован (и, следовательно, когда один ген orf6* был инактивирован, см. примеры 14 и 15), восстанавливается продуцирование спирамицина I. Это показывает, что ген orf5* необходим в биосинтезе спирамицинов, поскольку его инактивация вызывает полное прекращение продуцирования спирамицинов.
Ген orf5* кодирует протеин, обладающий относительно большим сходством с несколькими О-метилтрансферазами. Следовательно, ген orf5* будет являться О-метилтрансферазой, участвующей в биосинтезе платенолида. Для подтверждения этой гипотезы проводили эксперименты по анализу с помощью CL/SM и ЯМР при использовании штамма S. ambofaciens генотипа orf6*::attΩhyg+, полученного из штамма, сверхпродуцирующего спирамицины.
Образец супернатанта этого штамма приготовляли по способу, описанному выше (см. пример 16, без экстракции с помощью MIBK) и анализировали путем CL/SM, как описано выше (см. параграфы 20.2 и 20.3). Однако используемой колонкой является колонка X-Terra (Waters SAS, St-Quentin en-Yvelines, Франция) и конусное напряжение спектрометра установлено при 380 вольт для достижения фрагментации анализируемого соединения. В этих условиях был обнаружен продукт, время удерживания которого составляет примерно 13,1 минут. Масс-спектр этого соединения имеет вид, подобный таковому спирамицина I, но молекулярный ион имеет массу 829. Различие массы на 14 по отношению к массе спирамицина может объясняться отсутствием метильной группы у кислорода, который содержит углерод в положении 4 лактонового цикла (структура этого соединения представлена на фиг.39). Наличие соединения с массой 829 позволяет подтвердить гипотезу о роли orf5* в биосинтезе спирамицинов.
Путем микробиологического теста, осуществляемого при использовании чувствительного штамма M. luteus (см. пример 15 и фиг.18), было показано, что медиаторная молекула (спирамицин без метильной группы, структура которого представлена на фиг.39), продуцируемая штаммом orf6*::Ωatthyg+, намного менее активна (в 10 раз), чем исходный спирамицин с метильной группой в положении 4.
Пример 28
Конструирование новых “эксцизируемых кластеров”
Были сконструированы новые эксцизируемые кластеры. Эти кластеры очень похожи на эксцизируемые кластеры, уже описанные в примере 9. Основным различием между прежними и новыми кластерами является отсутствие в этих последних последовательностей, соответствующих концам интерпозона Ω, последовательностей, которые содержат терминатор транскрипции, происходящий от фага Т4.
В кластерах без терминатора ген, который придает резистентность к антибиотику, фланкирован последовательностями attR и attL, позволяющими осуществлять эксцизию. Геном резистентности является ген аас(3)IV, который кодирует ацетилтрансферазу, придающую резистентность к апрамицину. Этот ген находится в кластере Ωаас (номер доступа в банк генов: Х99313; Blondelet-Rouault M.H. и др., 1997) и был амплифицирован путем PCR при использовании, в качестве матрицы, плазмиды pOSK1102 (см. выше) и, в качестве праймеров, олигонуклеотидов KF42 и KF43, содержащих, каждый, сайт рестрикции HindIII (выделено жирным шрифтом) (AAGCTT) в 5'.
KF42: 5'-AAGCTTGTACGGCCCACAGAATGATGTCAC-3' (SEQ ID № 153) и
KF43: 5'-AAGCTTCGACTACCTTGGTGATCTCGCCTT-3' (SEQ ID № 154).
Полученный продукт PCR длиной примерно 1 т.н. клонировали в векторе E. coli pGEMT Easy с получением плазмиды pSPM83.
Вектор pSPM83 рестриктировали ферментом рестрикции HindIII. Фрагмент HindIII-HindIII вставки выделяли путем очистки из 0,8%-ного геля агарозы, затем клонировали в сайте HindIII, расположенном между последовательностями attL и attR различных плазмид, несущих различные возможные эксцизируемые кластеры (см. пример 9 и фиг.27), для замены фрагмента HindIII, соответствующего Ωacc, фрагментом HindIII, соответствующим одному гену аас. Это позволило получить кластеры att1aac, att2aac и att3aac (согласно желательной рамке считывания, см. пример 9). В зависимости от ориентации гена аас по отношению к последовательностям attL и attR различают att1aac+, att1aac-, att2aac+, att2aac-, att3aac+ и att3aac- (согласно тем же условиям, что и таковые, выбранные в примере 9).
Пример 29
Конструирование штамма S. ambofaciens, прерванного в гене orf28c
Инактивацию гена orf28c осуществляли по способу с использованием эксцизируемых кластеров. Эксцизируемый кластер att3aac+ (см. пример 28) амплифицировали путем PCR при использовании в качестве матрицы плазмиды pSPM101 (плазмида pAPM101 представляет собой плазмиду, происходящую от вектора pGP704Not (Chaveroche и др., 2000; Miller V.L. & Mekalanos J.J., 1988), в которой кластер aat3taac+ был клонирован в качестве фрагмента EcoRV в единственном сайте EcoRV pGP704Not) и с помощью следующих праймеров:
KF32:
5' CAACCGCTTGAGCTGCTCCATCAACTGCTGGGCCGAGGTATCGCGCGCGCTTCGTTCGGGACGAA 3' (SEQ ID № 155) и
KF33:
5' TGGGTCCCGCCGCGCGGCACGACTTCGACTCGCTCGTCTATCTGCCTCTTCGTCCCGAAGCAACT 3' (SEQ ID № 156)
39 Нуклеотидов, расположенных в конце 5' этих олигонуклеотидов, включают последовательность, соответствующую последовательности в гене orf28c, и 26 нуклеотидов, расположенных полностью в 3' (указано выше жирным шрифтом и подчеркнуто), соответствуют последовательности одного из концов эксцизируемого кластера att3aac+.
Таким образом полученный продукт PCR использовали для трансформации гиперрекомбинантного штамма E. coli DY330 (Yu и др., 2000) (этот штамм содержит гены exo, bet и gam фага лямбда, интегрированные в его хромосому, эти гены экспрессированы при температуре 42°С; этот штамм использовали вместо штамма E. coli KS272 (Chaveroche и др., 2000)), содержащего космиду pSPM36. Таким образом, бактерии трансформировали путем электропорации с помощью этого продукта PCR и клоны селекционировали по их резистентности к апрамицину. Космиды полученных клонов эктрагировали и рестриктировали с помощью фермента рестрикции BamHI с целью подтверждения того, что полученный профиль рестрикции соответствует ожидаемому профилю, если имела место инсерция кластера (att3aac+) в ген orf28c, то есть, если действительно имела место гомологичная рекомбинация между концами продукта PCR и гена-мишени. Подтверждение конструкции также может быть реализовано любым способом, известным специалисту, и, в частности, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR. Был отобран клон, космида которого обладает ожидаемым профилем, и соответствующая космида была названа pSPM107. Эта космида является производной pSPM36, в которой prf28c прерван кластером att3aac+. Инсерция кластера сопровождается делецией в гене orf28c, прерывание начинается на уровне 28-го кодона orf28c. После кластера остаются 137 последних кодонов orf28c.
Космиду pSPM107, во-первых, вводили в штамм E.coli DH5α, затем в штамм Streptomyces ambofaciens OSC2 путем трансформации протопластов. После трансформации клоны селекционировали по их резистентности к апрамицину. Резистентные к апрамицину клоны затем пересевали, соответственно, на среду с апрамицином (антибиотик В) и на среду с пуромицином (антибиотик А) (см. фиг.9). Клоны, резистентные к апрамицину (ApraR) и чувствительные к пиромицину (PuroS), представляют собой, в принципе, такие, где имел место двойной кроссинговер и которые обладают геном orf28c, прерванным кластером att3acc+. Эти клоны предпочтительно отбирали и замена исходной копии orf28c копией, прерванной кластером, была подтверждена путем гибридизации. Так, полную ДНК полученных клонов рестриктировали с помощью нескольких ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом, соответствующим кластеру att3aac+, для подтверждения наличия кластера в ожидаемом локусе в геномной ДНК полученных клонов. Подтверждение генотипа также может быть осуществлено любым способом, известным специалисту, и, в частности, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR.
В особенности был отобран клон, обладающий ожидаемыми характеристиками (orf28c::att3aac+), и назван SPM107. Этот клон, следовательно, имеет генотип orf28c::att3aac+ и был назван SPM107. Не нужно осуществлять эксцизию кластера для исследования воздействия инактивации orf28c, учитывая ориентацию генов (см. фиг.3). На самом деле, тот факт, что orf29 ориентирован в направлении, противоположном orf28c, показывает, что эти гены не являются сотранскрибированными. Использование эксцизируемого кластера, взамен, позволяет иметь возможность в любое время удалять маркер селекции, в частности, путем трансформации с помощью плазмиды pOSV508.
Для определения “гасящего” воздействия гена orf28c на продуцирование спирамицинов, продуцирование спирамицинов штаммом SPM107 было тестировано по способу, описанному в примере 15. Таким образом, показано, что этот штамм обладает фенотипом, не являющимся продуцентом спирамицинов. Это показывает, что ген orf28c представляет собой ген, существенный в биосинтезе спирамицина в S. ambofaciens.
Пример 30
Конструирование штамма S. ambofaciens, прерванного в гене orf31
Инактивацию гена orf31 осуществляли по способу с использованием эксцизируемых кластеров. Эксцизируемый кластер att3aac+ амплифицировали путем PCR при использовании, в качестве матрицы, плазмиды pSPM101 и олигонуклеотидов EDR71 и EDR72:
5' CGTCATCGACGTGCGGGGAAGACAGAGGTGATACCGATGATCGCGCGCGCTTCGTTCGGGACGAA 3' (SEQ ID № 157);
EDR72:
5' GCCAGCACCTCGTCCAGCTGCTCGACGGAACTCACCCCC ATCTGCCTCTTCGTCCCGAAGCAACT 3' (SEQ ID № 158)
39 Нуклеотидов, расположенных в конце 5' этих олигонуклеотидов, включают последовательность, соответствующую последовательности в гене orf31, и 26 нуклеотидов, расположенных полностью в 3' (указано выше жирным шрифтом и подчеркнуто), соответствуют последовательности одного из концов эксцизируемого кластера att3aac+.
Таким образом полученный продукт PCR использовали для трансформации штамма E. coli KS272, содержащего плазмиду pKOBEG и космиду pSPM36, как описано Chaveroche и др. (Chaveroche и др., 2000) (см. фиг.12 по принципу: плазмида pOS49.99 должна быть заменена космидой pSPM36 и полученная плазмида не является больше плазмидой pSPM17, а представляет собой плазмиду pSPM543). Таким образом, бактерии трансформировали путем электропорации с помощью этого продукта PCR и клоны селекционировали по их резистентности к апрамицину. Космиды полученных клонов экстрагировали и рестриктировали с помощью нескольких ферментов рестрикции с целью подтверждения того, что полученный профиль рестрикции соответствует ожидаемому профилю, если имела место инсерция кластера (att3aac+) в ген orf31, то есть, если действительно имела место гомологичная рекомбинация между концами продукта PCR и гена-мишени. Подтверждение конструкции также может быть реализовано любым способом, известным специалисту, и, в частности, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR. Был отобран клон, космида которого обладает ожидаемым профилем, и соответствующая космида была названа pSPM543. Эта космида является производной pSPM36, в которой prf31 прерван кластером att3aac+ (см. фиг.12). Инсерция кластера сопровождается делецией в гене orf31, прерывание начинается на уровне тридцать шестого кодона orf31. После кластера остаются 33 последних кодона orf31.
Космиду pSPM543 вводили в штамм Streptomyces ambofaciens OSC2 (см. выше) путем трансформации протопластов (Kieser T. и др., 2000). После трансформации клоны селекционировали по их резистентности к апрамицину. Резистентные к апрамицину клоны затем пересевали, соответственно, на среду с апрамицином (антибиотик В) и на среду с пуромицином (антибиотик А) (см. фиг.9). Клоны, резистентные к апрамицину (ApraR) и чувствительные к пиромицину (PuroS), представляют собой, в принципе, такие, где имел место двойной кроссинговер и которые обладают геном orf31, прерванным кластером att3acc+. Эти клоны предпочтительно отбирали и замена исходной копии orf31 копией, прерванной кластером, была подтверждена путем гибридизации. Так, полную ДНК полученных клонов рестриктировали с помощью нескольких ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом, соответствующим кластеру att3aac+, для подтверждения наличия кластера в ожидаемом локусе в геномной ДНК полученных клонов. Вторую гибридизацию осуществляли при использовании в качестве зонда фрагмента ДНК, полученного путем PCR и содержащего очень большую область кодирующей последовательности гена orf31.
Подтверждение генотипа также может быть осуществлено любым способом, известным специалисту, и, в частности, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR.
Предпочтительно был отобран клон, обладающий ожидаемыми характеристиками (orf31::att3aac+), и назван SPM543. В самом деле, благодаря двум гибридизациям было подтверждено, что кластер att3aac+ действительно находится в геноме этого клона и что действительно получают ожидаемый профиль рестрикции в случае замены, за счет двойной рекомбинации, исходного гена копией, прерванной кластером att3aac+ в геноме этого клона. Этот клон, следовательно, имеет генотип orf31::att3aac+ и был назван SPM543. Не нужно осуществлять эксцизию кластера для исследования воздействия инактивации orf31, учитывая ориентацию генов (см. фиг.3). В самом деле, тот факт, что orf32c ориентирован в направлении, противоположном orf31, показывает, что эти гены не являются сотранскрибированными. Использование эксцизируемого кластера, взамен, позволяет иметь возможность в любое время удалять маркер селекции, в частности, путем трансформации с помощью плазмиды pOSV508.
Для определения “гасящего” воздействия гена orf31 на продуцирование спирамицинов, продуцирование спирамицинов штаммом SPM543 было тестировано по способу, описанному в примере 15. Таким образом, показано, что этот штамм обладает фенотипом, не являющимся продуцентом спирамицинов. Это показывает, что ген orf31 представляет собой ген, существенный в биосинтезе спирамицина в S. ambofaciens.
Пример 31
Конструирование штамма S. ambofaciens, прерванного в гене orf32c
Инактивацию гена orf32c осуществляли по способу с использованием эксцизируемых кластеров. Эксцизируемый кластер att3aac+ амплифицировали путем PCR при использовании в качестве матрицы плазмиды pSPM101 и с помощью следующих праймеров:
KF52:
5' GATCCGCCAGCCTCACGTCACGCCGCGCCGCCTCCCTGAC ATCGCGCGCGCTTCGTTCGGGACGAA 3' (SEQ ID № 159) и
KF53:
5' GAGGCGGACGTCGGTACGCGGTGGGAGCCGGAGTTCGACA ATCTGCCTCTTCGTCCCGAAGCAACT 3' (SEQ ID № 160).
40 Нуклеотидов, расположенных в конце 5' этих олигонуклеотидов, включают последовательность, соответствующую последовательности в гене orf32c, и 26 нуклеотидов, расположенных полностью в 3' (указано выше жирным шрифтом и подчеркнуто), соответствуют последовательности одного из концов эксцизируемого кластера att3aac+.
Таким образом полученный продукт PCR использовали для трансформации гиперрекомбинантного штамма E. coli DY330 (Yu и др., 2000), содержащего космиду pSPM36. Таким образом, бактерии трансформировали путем электропорации с помощью этого продукта PCR и клоны селекционировали по их резистентности к апрамицину. Космиды полученных клонов эктрагировали и рестриктировали с помощью фермента рестрикции BamHI с целью подтверждения того, что полученный профиль рестрикции соответствует ожидаемому профилю, если имела место инсерция кластера (att3aac+) в ген orf32c, то есть, если действительно имела место гомологичная рекомбинация между концами продукта PCR и гена-мишени. Подтверждение конструкции также может быть реализовано любым способом, известным специалисту, и, в частности, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR. Был отобран клон, космида которого обладает ожидаемым профилем, и соответствующая космида была названа pSPM106. Эта космида происходит от pSPM36, в которой prf32c прерван кластером att3aac+. Инсерция кластера сопровождается делецией в гене orf32c, прерывание начинается на уровне 112-го кодона orf31c. После кластера остаются 91 последних кодонов orf32c.
Космиду pSPM106, во-первых, вводили в штамм E.coli DH5α, затем в штамм Streptomyces ambofaciens OSC2 путем трансформации. После трансформации клоны селекционировали по их резистентности к апрамицину. Резистентные к апрамицину клоны затем пересевали, соответственно, на среду с апрамицином (антибиотик В) и на среду с пуромицином (антибиотик А) (см. фиг.9). Клоны, резистентные к апрамицину (ApraR) и чувствительные к пуромицину (PuroS), представляют собой, в принципе, такие, где имел место двойной кроссинговер и которые обладают геном orf32c, прерванным кластером att3acc+. Эти клоны предпочтительно отбирали и замена исходной копии orf32c копией, прерванной кластером, была подтверждена путем гибридизации. Так, полную ДНК полученных клонов рестриктировали с помощью нескольких ферментов, разделяли на геле агарозы, переносили на мембрану и гибридизировали с зондом, соответствующим кластеру att3aac+, для подтверждения наличия кластера в геномной ДНК полученных клонов. Подтверждение генотипа также может быть осуществлено любым способом, известным специалисту, и, в частности, путем PCR при использовании соответствующих олигонуклеотидов и секвенирования продукта PCR.
В особенности был отобран клон, обладающий ожидаемыми характеристиками (orf32c::att3aac+). Этот клон, следовательно, имеет генотип orf32c::att3aac+ и был назван SPM106. Не нужно осуществлять эксцизию кластера для исследования воздействия инактивации orf32c, учитывая ориентацию генов (см. фиг.3). На самом деле, тот факт, что orf33 ориентирован в направлении, противоположном orf32c, показывает, что эти гены не являются сотранскрибированными. Использование эксцизируемого кластера, взамен, позволяет иметь возможность в любое время удалять маркер селекции, в частности, путем трансформации с помощью плазмиды pOSV508.
Для определения “гасящего” воздействия гена orf32c на продуцирование спирамицинов, продуцирование спирамицинов штаммом SPM106 было тестировано по способу, описанному в примере 15. Таким образом, показано, что этот штамм обладает фенотипом, не являющимся продуцентом спирамицинов. Это показывает, что ген orf32c представляет собой ген, существенный в биосинтезе спирамицина в S. ambofaciens.
Список конструкций, описанных в настоящей заявке
Список аббревиатур: Am: ампициллин; Hyg: гигромицин; Sp: спирамицин; Ts: тиострептон; Cm: хлорамфеникол; Kn: канамицин; Apra: апрамицин.
Депонирование биологического материала
Следующие организмы были депонированы 10 июля 2002 г. в Национальной коллекции культур микроорганизмов (CNCM), 25 rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, согласно положениям Будапештского Соглашения.
- Штамм OSC2 под регистрационным номером I-2908.
- Штамм SPM501 под регистрационным номером I-2909.
- Штамм SPM502 под регистрационным номером I-2910.
- Штамм SPM507 под регистрационным номером I-2911.
- Штамм SPM508 под регистрационным номером I-2912.
- Штамм SPM509 под регистрационным номером I-2913.
- Штамм SPM21 под регистрационным номером I-2914.
- Штамм SPM22 под регистрационным номером I-2915.
- Штамм OS49.67 под регистрационным номером I-2916.
- Штамм OS49.107 под регистрационным номером I-2917.
- Штамм Escherichia coli DH5α, содержащий плазмиду pOS44.4, под регистрационным номером I-2918.
Следующие организмы были депонированы 26 февраля 2003 г. в Национальной коллекции культур микроорганизмов (CNCM), 25 rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, согласно положениям Будапештского Соглашения.
- Штамм SPM502pSPM525 под регистрационным номером I-2977.
Следующие организмы были депонированы в Национальной коллекции культур микроорганизмов (CNCM), 25 rue du Docteur Roux, 75724 Париж, Cedex 15, Франция, 6 октября 2003 г., согласно положениям Будапештского Соглашения.
- Штамм OSC2/pSPM75(2) под регистрационным номером I-3101.
Все цитированные публикации и патенты включены в настоящую заявку путем ссылки.
Библиография:
- Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. J Mol Biol. (1990). 215 (3):403-410.
- Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res. (1997). 25 (17):3389-402.
- Amann,E., Ochs,B. и Abel,K.J. Tightly regulated tac promoter vectors useful for the expression of unfused and fused proteins in Escherichia coli Gene (1988) 69 (2), 301-315.
- Arisawa,A., Kawamura,N., Tsunekawa,H., Okamura,K., Tone,H. и Okamoto,R. Cloning and nucleotide sequences of two genes involved in the 4''-O-acylation of macrolide antibiotics from Streptomyces thermotolerans. Biosci. Biotechnol. Biochem. (1993). 57 (12) : 2020-2025.
- Arisawa A, Kawamura N, Takeda K, Tsunekawa H, Okamura K, Okamoto R. Cloning of the macrolide antibiotic biosynthesis gene acyA, which encodes 3-O-acyltransferase, from Streptomyces thermotolerans and its use for direct fermentative production of a hybrid macrolide antibiotic. Appl Environ Microbiol. (1994). 60 (7):2657-2660.
- August,P.R., Tang,L., Yoon,Y.J., Ning,Streptomyces, Mueller,R., Yu,T.W., Taylor,M., Hoffmann,D., Kim,C.G., Zhang,X., Hutchinson,C.R. и Floss,H.G. Biosynthesis of the ansamycin antibiotic rifamycin: deductions from the molecular analysis of the rif biosynthetic gene cluster of Amycolatopsis mediterranei S699. Chem. Biol. (1998) 5 (2), 69-79.
- Ausubel Fred M., Brent Roger, Kingston Robert E., Moore David D., Seidman J.G., Smith John A., Struhl Kevin (Редакторы). Current Protocols in Molecular Biology, опубликованный John Wiley & Sons Inc. Current Protocols Customer Service, 605 Third Avenue, 9th Floor New York, NY 10158 USA (выпуск: март 2002).
- Baltz RH, McHenney MA, Cantwell CA, Queener SW, Solenberg PJ. Applications of transposition mutagenesis in antibiotic producing streptomycetes. Antonie Van Leeuwenhoek. (1997). 71 (1-2): 179-187.
- Bate N, Butler AR, Gandecha AR, Cundliffe E. Multiple regulatory genes in the tylosin biosynthetic cluster of Streptomyces fradiae. Chem Biol. (1999). 6 (9): 617-624.
- Bate,N., Butler,A.R., Smith,I.P. и Cundliffe,E. The mycarose-biosynthetic genes of Streptomyces fradiae, producer of tylosin. Microbiology (2000). 146 (Pt 1), 139-146.
- Bayley C., Morgan, Dale E. C., Ow D. W. Exchange of gene activity in transgenic plants catalysed by the Cre-lox site specific system. Plant Mol. Biol (1992). 18 :353-361.
- Bentley,S.D., Chater,K.F., Cerdeno-Tarraga,A.M., Challis,G.L., Thomson,N.R., James,K.D., Harris,D.E., Quail,M.A., Kieser,H., Harper,D., Bateman,A., Brown,S., Chandra,G., Chen,C.W., Collins,M., Cronin,A., Fraser,A., Goble,A., Hidalgo,J., Hornsby,T., Howarth,S., Huang,C.H., Kieser,T., Larke,L., Murphy,L., Oliver,K., O'Neil,S., Rabbinowitsch,E., Rajandream,M.A., Rutherford,K., Rutter,S., Seeger,K., Saunders,D., Sharp,S., Squares,R., Squares,S., Taylor,K., Warren,T., Wietzorrek,A., Woodward,J., Barrell,B.G., Parkhill,J. и Hopwood,D.A. Complete genome sequence of the model actinomycete Streptomyces coelicolor A3(2). Nature (2002). 417 (6885), 141-147.
- Bibb MJ, Findlay PR, Johnson MW. The relationship between base composition and codon usage in bacterial genes and its use for the simple and reliable identification of protein-coding sequences. Gene. (1984) 30 (1-3):157-166.
- Bibb MJ, Janssen GR, Ward JM. Cloning and analysis of the promoter region of the erythromycin resistance gene (ermE) of Streptomyces erythraeus. Gene. (1985). 38 (1-3) : 215-26.
- Bibb MJ, White J, Ward JM, Janssen GR. The mRNA for the 23S rRNA methylase encoded by the ermE gene of Saccharopolyspora erythraea is translated in the absence of a conventional ribosome-binding site. Mol Microbiol. (1994). 14 (3): 533-45.
- Bierman M, Logan R, O'Brien K, Seno ET, Rao RN, Schoner BE. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp. Gene. 1992;116(1):43-9.
- Blondelet-Rouault M-H., Weiser J., Lebrihi A., Branny P. и Pernodet J-L. Antibiotic resistance gene cassettes derived from the Ω interposon for use in E. coli and Streptomyces. Gene (1997) 190 : 315-317.
- Boccard F., Smokvina T., J.-L., Pernodet Fridmann A., и Guérineau M., Structural analysis of loci involved in pSAM2 site-specific integration in Streptomyces. Plasmid, (1989a). 21 : 59-70.
- Boccard F., Smokvina T., J.-L., Pernodet Fridmann A. и Guérineau M.. The integrated conjugative plasmpid pSAM2 of Streptomyces ambofaciens is related to temperate bacteriophages. EMBO J. (1989b) 8 : 973-980.
- Brunelli J.P., Pall M.L., A series of Yeast/ E. coli lambda expression vectors designed for directional cloning of cDNA and cre/lox mediated plasmid excision. Yeast. (1993) 9 : 1309-1318.
- Camilli A., Beattie D. T. и Mekalanos J. J. Use of genetic recombination as a reporter of gene expression. Proc. Natl. Acad. Sci. USA (1994) 91(7), 2634-2638.
- Campelo,A.B. и Gil,J.A. The candicidin gene cluster from Streptomyces griseus IMRU 3570. Microbiology (2002) 148 (Pt 1), 51-59.
- Carreras C, Frykman S, Ou S, Cadapan L, Zavala S, Woo E, Leaf T, Carney J, Burlingame M, Patel S, Ashley G, Licari P. Saccharopolyspora erythraea-catalyzed bioconversion of 6-deoxyerythronolide B analogs for production of novel erythromycins. J Biotechnol. (2002). 92(3): 217-28.
- Chao K.-M., Pearson W.R., Miller W. Aligning two sequences within a specified diagonal band. Comput. Appl. Biosci. (1992) 8 : 481-487.
- Chater K. F. The improving prospects for yield increase by genetic engineering in antibiotic-producing Streptomycetes. Biotechnology. (1990). 8 (2) : 115-121.
- Chaveroche MK, Ghigo JM, d'Enfert C. A rapid method for efficient gene replacement in the filamentous fungus Aspergillus nidulans. Nucleic Acids Res. 2000; 28(22) :E97.
- Church GM, Gilbert W. Genomic sequencing. Proc Natl Acad Sci U S A. (1984) 81 (7):1991-5.
- Churchward G, Belin D, Nagamine Y. A pSC101-derived plasmid which shows no sequence homology to other commonly used cloning vectors. Gene. (1984) 31 (1-3): 165-71.
- Comstock, L.E., Coyne,M.J., Tzianabos,A.O. и Kasper,D.L. Interstrain variation of the polysaccharide B biosynthesis locus of Bacteroides fragilis: characterization of the region from strain 638R J. Bacteriol. (1999). 181 (19) : 6192-6196.
- Cox KL, Baltz RH. Restriction of bacteriophage plaque formation in Streptomyces spp. J Bacteriol. (1984) 159(2): 499-504.
- Dale E.C., Ow D.W., Gene transfer with subsequent removal of the selection gene from the host genome. Proc. Natl. Acad. Sci. USA (1991). 88 : 10558-10562.
- Doumith M, Weingarten P, Wehmeier UF, Salah-Bey K, Benhamou B, Capdevila C, Michel JM, Piepersberg W, Raynal MC. Analysis of genes involved in 6-deoxyhexose biosynthesis and transfer in Saccharopolyspora erythraea. Mol Gen Genet. (2000) 264(4): 477-485.
- Draeger, G., Park, Streptomyces-H.H. и Floss,H.G. Mechanism of the 2-deoxygenation step in the biosynthesis of the deoxyhexose moieties of the antibiotics granaticin and oleandomycin J. Am. Chem. Soc. (1999) 121 : 2611-2612.
- Gandecha, A.R., Large,S.L. и Cundliffe,E. Analysis of four tylosin biosynthetic genes from the tylLM region of the Streptomyces fradiae genome. Gene. (1997). 184 (2) :197-203.
- Geistlich,M., Losick,R., Turner,J.R. и Rao,R.N. Characterization of a novel regulatory gene governing the expression of a polyketide synthase gene in Streptomyces ambofaciens. Mol. Microbiol. (1992). 6 (14) : 2019-2029.
- Gourmelen A, Blondelet-Rouault MH, Pernodet JL. Characterization of a glycosyl transferase inactivating macrolides, encoded by gimA from Streptomyces ambofaciens. Antimicrob Agents Chemother. (1998). 42(10): 2612-9.
- Huang X. и Miller W. A time-efficient, linear-space local similarity algorithm. Adv. Appl. Math. (1991). 12 : 337-357.
- Hara O и Hutchinson CR. Cloning of midecamycin(MLS)-resistance genes from Streptomyces mycarofaciens, Streptomyces lividans and Streptomyces coelicolor A3(2). J Antibiot (Tokyo). (1990). 43 (8):977-991.
- Hara O и Hutchinson CR. A macrolide 3-O-acyltransferase gene from the midecamycin-producing species Streptomyces mycarofaciens. J Bacteriol. (1992). 174(15) :5141-5144.
- Hoffmeister D, Ichinose K, Domann S, Faust B, Trefzer A, Drager G, Kirschning A, Fischer C, Kunzel E, Bearden D, Rohr J и Bechthold A. The NDP-sugar co-substrate concentration and the enzyme expression level influence the substrate specificity of glycosyltransferases: cloning and characterization of deoxysugar biosynthetic genes of the urdamycin biosynthetic gene cluster. Chem Biol. (2000). 7 (11):821-831.
- Hopwood, D.A. Future possibilities for the discovery of new antibiotics by genetic engineering. In Beta-lactam antibiotics. Под редакцией M.R.J. Salton и G.D. Shockman. New York: Academic Press. (1981). 585-598.
- Hopwood D. A., Malpartida F., Kieser H. M., Ikeda H., Duncan J., Fujii I., Rudd A. M., Floss H. G. и Omura S. Production of 'hybrid' antibiotics by genetic engineering. Nature. (1985a). 314 (6012): 642-644.
- Hopwood D. A., Malpartida F., Kieser H. M., Ikeda H. и Omura S. (1985b) In Microbiology (ed S. Silver). American Society for Microbiology, Washington D. C., 409-413.
- Houben Weyl, 1974, d Meuthode der Organischen Chemie, под редакцией E. Wunsch Ed., том 15-I и 15-II,
- Hutchinson, C.R. Prospects for the discovery of new (hybrid) antibiotics by genetic engineering of antibiotic-producing bacteria. Med Res Rev. (1988). 8 (4) : 557-567.
- Hutchinson C. R., Borell C. W., Otten S. L., Stutzman-Engwall K. J. и Wang Y. Drug discovery and development through the genetic engineering of antibiotic-producing microorganisms J. Med. Chem. (1989) 32 (5) : 929-937.
- Ishikawa J, Hotta K. FramePlot: a new implementation of the frame analysis for predicting protein-coding regions in bacterial DNA with a high G + C content. FEMS Microbiol Lett. (1999) 174(2):251-3.
- Kieser, T, Bibb, MJ, Buttner MJ, Chater KF, Hopwood DA. Practical Streptomyces Genetics. 2000. The John Innes Foundation, Norwich UK.
- Kuhstoss и др. Production of a novel polyketide through the construction of a hybrid polyketide synthase. Gene. (1996). 183(1-2): 231-236.
- Lacalle RA, Pulido D, Vara J, Zalacain M, Jimenez A. Molecular analysis of the pac gene encoding a puromycin N-acetyl transferase from Streptomyces alboniger. Gene. (1989). 79(2):375-380.
- Lakso M, Sauer B, Mosinger B Jr, Lee EJ, Manning RW, Yu SH, Mulder KL, Westphal H. Targeted oncogene activation by site-specific recombination in transgenic mice. Proc Natl Acad Sci U S A. (1992) 89 (14) :6232-6.
- Li,T.B., Shang,G.D., Xia,H.Z. и Wang,Y.G. Cloning of the sugar related biosynthesis gene cluster from Streptomyces tenebrarius H6. Sheng Wu Gong Cheng Xue Bao (2001). 17 (3) : 329-331.
- Ligon,J., Hill,S., Beck,J., Zirkle,R., Molnar,I., Zawodny,J., Money,S. и Schupp,T. Characterization of the biosynthetic gene cluster for the antifungal polyketide soraphen A from Sorangium cellulosum So ce26. Gene (2002). 285 (1-2), 257-267.
- Liu L, Saevels J, Louis P, Nelis H, Rico S, Dierick K, Guyomard S, Roets E, Hoogmartens J. Interlaboratory study comparing the microbiological potency of spiramycins I, II and III. J Pharm Biomed Anal. (1999). 20 (1-2):217-24.
- Mazodier P, Petter R, Thompson C. Intergeneric conjugation between Escherichia coli and Streptomyces species.J Bacteriol. (1989). 171 (6):3583-5.
- Merrifield RB, 1965a, Nature, 207(996): 522-523.
- Merrifield RB., 1965b, Science, 150(693): 178-185.
- Merson-Davies,L.A. и Cundliffe,E. Analysis of five tylosin biosynthetic genes from the tyllBA region of the Streptomyces fradiae genome, Molecular microbiology. (1994). 13 (2) : 349-355.
- Miller VL, Mekalanos JJ. A novel suicide vector and its use in construction of insertion mutations: osmoregulation of outer membrane proteins and virulence determinants in Vibrio cholerae requires toxR. J Bacteriol. (1988) 170(6):2575-83.
- Nakano, Y., Yoshida, Y., Yamashita, Y. & Koga, T. Construction of a series of pACYC-derived plasmid vectors. Gene (1995). 162 : 157-8.
- Nielsen H., Engelbrecht J., Brunak S. и von Heijne G. Identification of prokaryotic and eukaryotic signal peptides and prediction of their cleavage sites. Protein Engineering. (1997). 10: 1-6.
- Oh SH, Chater KF. Denaturation of circular or linear DNA facilitates targeted integrative transformation of Streptomyces coelicolor A3(2): possible relevance to other organisms. J Bacteriol. 1997; 179 (1):122-7.
- Olano C, Lomovskaya N, Fonstein L, Roll JT, Hutchinson CR. A two-plasmid system for the glycosylation of polyketide antibiotics: bioconversion of epsilon-rhodomycinone to rhodomycin D. Chem Biol. (1999). 6 (12):845-55.
- Omura S, Kitao C, Hamada H, Ikeda H. Bioconversion and biosynthesis of 16-membered macrolide antibiotics. X. Final steps in the biosynthesis of spiramycin, using enzyme inhibitor: cerulenin. Chem Pharm Bull (Tokyo). (1979a). 27(1): 176-82.
- Omura S, Ikeda H, Kitao C. Isolation and properties of spiramycin I 3-hydroxyl acylase from Streptomyces and ambofaciens. J Biochem (Tokyo). (1979b). 86(6): 1753-8.
- Omura,S., Ikeda,H., Ishikawa,J., Hanamoto,A., Takahashi,C., Shinose,M., Takahashi,Y., Horikawa,H., Nakazawa,H., Osonoe,T., Kikuchi,H., Shiba,T., Sakaki,Y. и Hattori,M. Genome sequence of an industrial microorganism Streptomyces avermitilis: Deducing the ability of producing secondary metabolites. Proc. Natl. Acad. Sci. U.S.A. (2001). 98 (21), 12215-12220.
- Pearson W.R. и D. J. Lipman. Improved Tools for Biological Sequence Analysis. Proc. Natl. Acad. Sci. USA 1988, 85: 2444-2448.
- Pearson W. R. Rapid and Sensitive Sequence Comparison with FASTP and FASTA. Methods in Enzymology. 1990, 183: 63-98.
- Pernodet JL, Simonet JM, Guerineau M. Plasmids in different strains of Streptomyces ambofaciens: free and integrated form of plasmid pSAM2. Mol Gen Genet. (1984);198 (1):35-41.
- Pernodet JL, Alegre MT, Blondelet-Rouault MH, Guerineau M. Resistance to spiramycin in Streptomyces ambofaciens, the producer organism, involves at least two different mechanisms. J Gen Microbiol. (1993) ; 139 ( Pt 5):1003-11.
- Pernodet JL, Fish S, Blondelet-Rouault MH, Cundliffe E. The macrolide-lincosamide-streptogramin B resistance phenotypes characterized by using a specifically deleted, antibiotic-sensitive strain of Streptomyces lividans. Antimicrob Agents Chemother. (1996), 40 (3): 581-5.
- Pernodet JL, Gourmelen A, Blondelet-Rouault MH, Cundliffe E. Dispensable ribosomal resistance to spiramycin conferred by srmA in the spiramycin producer Streptomyces ambofaciens. Microbiology. (1999), 145 (Pt 9):2355-64.
- Pfoestl,A., Hofinger,A., Kosma,P. и Messner,P. Biosynthesis of dTDP-3-acetamido-3,6-dideoxy-alpha-D-galactose in Aneurinibacillus thermoaerophilus L420-91T. J. Biol. Chem. (2003), 278 (29): 26410-26417.
- Rao RN, Richardson MA, Kuhstoss S. Cosmid shuttle vectors for cloning and analysis of Streptomyces DNA. Methods Enzymol. (1987); 153: 166-98.
- Raynal A, Tuphile K, Gerbaud C, Luther T, Guerineau M, и Pernodet JL.. Structure of the chromosomal insertion site for pSAM2: functional analysis in Escherichia coli. Molecular Microbiology (1998) 28 (2) 333-342.
- Redenbach,M., Kieser,H.M., Denapaite,D., Eichner,A., Cullum,J., Kinashi,H. и Hopwood,D.A. A set of ordered cosmids and a detailed genetic and physical map for the 8 Mb Streptomyces coelicolor A3(2) chromosome Mol. Microbiol. (1996). 21 (1) : 77-96.
- Richardson MA, Kuhstoss S, Solenberg P, Schaus NA, Rao RN. A new shuttle cosmid vector, pKC505, for streptomycetes: its use in the cloning of three different spiramycin-resistance genes from a Streptomyces ambofaciens library. Gene. (1987); 61 (3):231-41.
- Robinson, J.A. Enzymes of Secondary Metabolism in Microorganisms. Chem Soc Rev. (1988). 17 :383-452.
- Russell SH, Hoopes JL, Odell JT. Directed excision of a transgene from the plant genome. Mol Gen Genet. (1992). 234 (1): 49-59.
- Sambrook J, Frisch EF, Maniatis T . Molecular Cloning : a laboratory manual 2nd ed. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York USA (1989).
- Schoner B, Geistlich M, Rosteck P Jr, Rao RN, Seno E, Reynolds P, Cox K, Burgett S и Hershberger C. Sequence similarity between macrolide-resistance determinants and ATP-binding transport proteins. Gene. (1992). 115 (1-2), 93-96.
- Sezonov G, Hagege J, Pernodet JL, Friedmann A и Guerineau M. Characterization of pra, a gene for replication control in pSAM2, the integrating element of Streptomyces ambofaciens. Mol Microbiol. (1995). 17(3): 533-44.
- Sezonov G., Blanc V., Bamas-Jacques N., Friedmann A., Pernodet J.L. и M. Guerineau, Complete conversion of antibiotic precursor to pristinamycin IIA by over expression of Streptomyces pristinaespiralis biosynthetic genes, Nature Biotechnology. (1997) 15 : 349-353.
- Sezonov G, Possoz C, Friedmann A, Pernodet JL, Guerineau M. KorSA from the Streptomyces integrative element pSAM2 is a central transcriptional repressor: target genes and binding sites. J Bacteriol. (2000). 182(5):1243-50.
- Simon R., Priefer U и Pühler. A broad host range mobilisation system for in vivo genetic engeneering : transposon mutagenesis in gram negative bacteria. Bio/Technology (1983). 1 : 784-791.
- Simon, и др., Plasmid vectors for the genetic analysis and manipulation of rhizobia and other gram-negative bacteria, p. 640-659. In A. Weissbach, and H. Weissbach (eds.), Methods in enzymology, vol 118, Academic Press, Inc., Orlando, 1986
- Summers,R.G., Donadio,Streptomyces, Staver,M.J., Wendt-Pienkowski,E., Hutchinson,C.R. и Katz,L. Sequencing and mutagenesis of genes from the erythromycin biosynthetic gene cluster of Saccharopolyspora erythraea that are involved in L-mycarose and D-desosamine production. Microbiology (1997). 143 (Pt 10) : 3251-3262.
- Van Mellaert L, Mei L, Lammertyn E, Schacht S, Anne J. Site-specific integration of bacteriophage VWB genome into Streptomyces venezuelae and construction of a VWB-based integrative vector. Microbiology. (1998). 144 ( Pt 12): 3351-8.
- Vara J, Lewandowska-Skarbek M, Wang YG, Donadio S, Hutchinson CR.Cloning of genes governing the deoxysugar portion of the erythromycin biosynthesis pathway in Saccharopolyspora erythraea (Streptomyces erythreus). J Bacteriol. (1989). 171(11): 5872-81.
- Vara J, Malpartida F, Hopwood DA, Jimenez A. Cloning and expression of a puromycin N-acetyl transferase gene from Streptomyces alboniger in Streptomyces lividans and Escherichia coli. Gene. (1985). 33(2): 197-206.
- Wahl, G. M., K. A. Lewis, J. C. Ruiz, B. Rothenberg, J. Zhao и G. A. Evans. Cosmid vectors for rapid genomic walking, restriction mapping, and gene transfer. Proc. Natl. Acad. Sci. USA (1987). 84: 2160-2164.
- Waldron,C., Matsushima,P., Rosteck,P.R., Broughton,M.C., Turner,J., Madduri,K., Crawford,K.P., Merlo,D.J. и Baltz,R.H. Cloning and analysis of the spinosad biosynthetic gene cluster of Saccharopolyspora spinosa(1) Chem. Biol. (2001) 8 (5) : 487-499.
- Walczak RJ, Hines JV, Strohl WR, Priestley ND.Bioconversion of the anthracycline analogue desacetyladriamycin by recombinant DoxA, a P450-monooxygenase from Streptomyces sp. strain C5. Org Lett. (2001). 3 (15):2277-9.
- Wang,Z.X., Li,S.M. и Heide,L. Identification of the coumermycin A(1) biosynthetic gene cluster of Streptomyces rishiriensis DSM 40489. Antimicrob. Agents Chemother. (2000) 44 (11), 3040-3048.
- Ward, J.M., Janssen, G.R., Kieser, T, Bibb, M.J., Buttner, M.J. & Bibb, M.J. Construction and characterization of a series of multi-copy promoter-probe plasmid vectors for Streptomyces using the aminoglycoside phosphotransferase from Tn5 as indicator. Mol Gen Genet (1986). 203 : 468-478.
- Wehmeier UF. New multifunctional Escherichia coli-Streptomyces shuttle vectors allowing blue-white screening on XGal plates. Gene. (1995). 165(1): 149-150.
- Wilms B, Hauck A, Reuss M, Syldatk C, Mattes R, Siemann M, Altenbuchner J. High-cell-density fermentation for production of L-N-carbamoylase using an expression system based on the Escherichia coli rhaBAD promoter. Biotechnol Bioeng. (2001). 73(2) :95-103.
- Worley K. C., Wiese B. A. и Smith R. F. BEAUTY: An enhanced BLAST-based search tool that integrates multiple biological information resources into sequence similarity search results. Genome Research (1995). 5: 173-184.
- Wu K, Chung L, Revill WP, Katz L и Reeves CD. The FK520 gene cluster of Streptomyces hygroscopicus var. ascomyceticus (ATCC 14891) contains genes for biosynthesis of unusual polyketide extender units. Gene. (2000). 251 (1): 81-90.
- Xue Y, Zhao L, Liu HW и Sherman DH. A gene cluster for macrolide antibiotic biosynthesis in Streptomyces venezuelae: architecture of metabolic diversity. Proc Natl Acad Sci U S A. (1998). 95 (21): 12111-12116.
- Yu D, Ellis HM, Lee EC, Jenkins NA, Copeland NG и Court DL. An efficient recombination system for chromosome engineering in Escherichia coli. Proc Natl Acad Sci U S A. (2000). 97(11): 5978-83.























































































































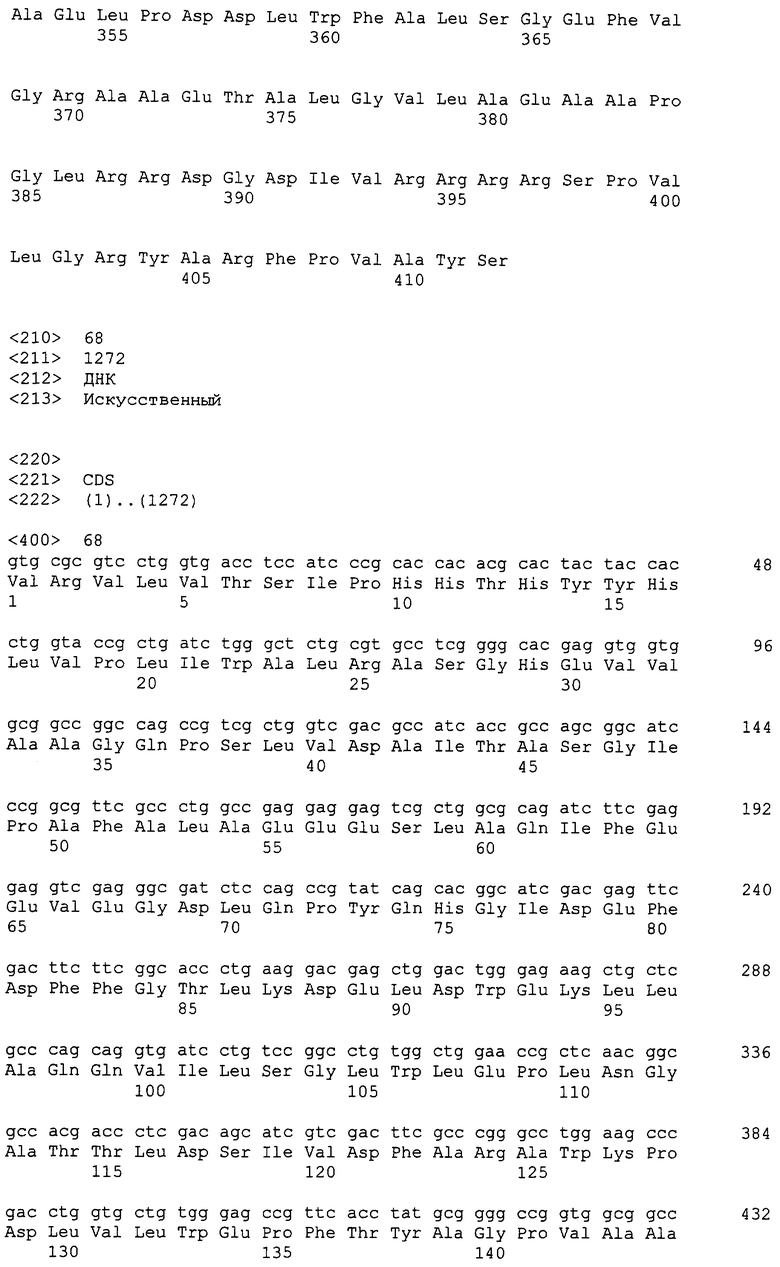







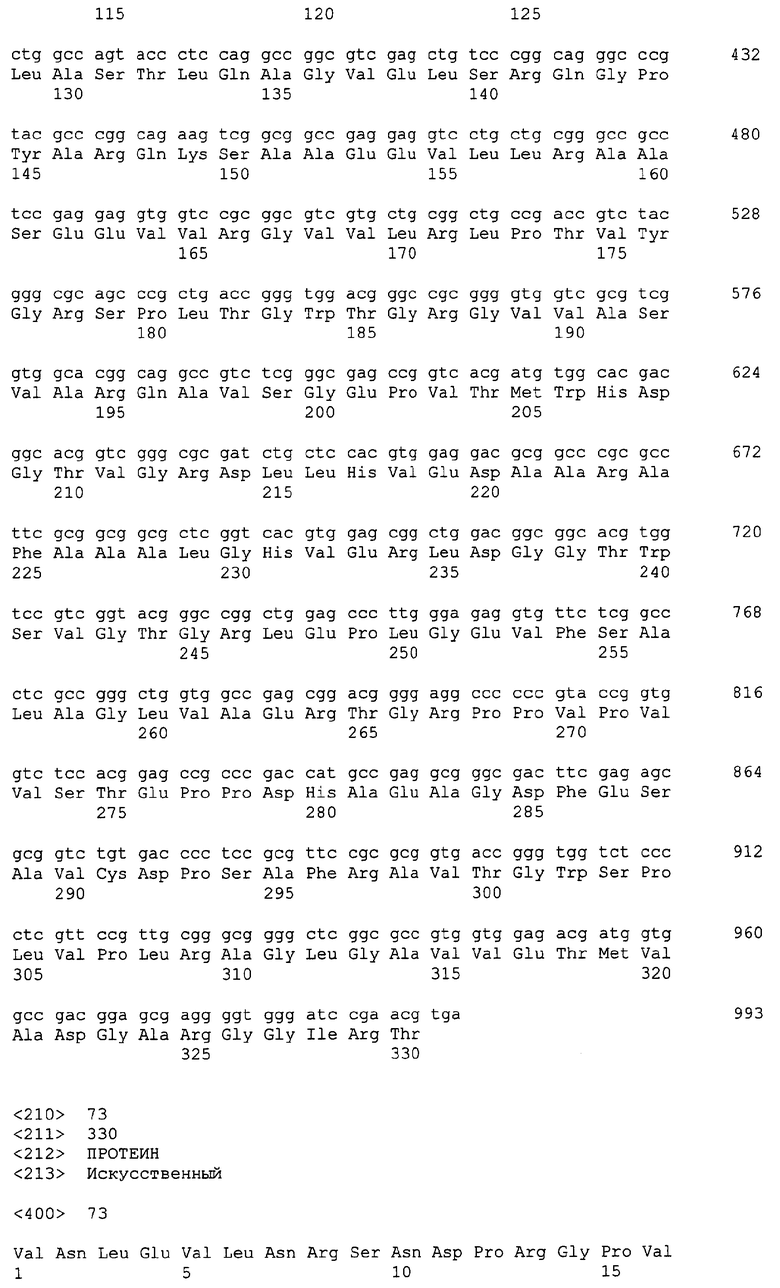























































































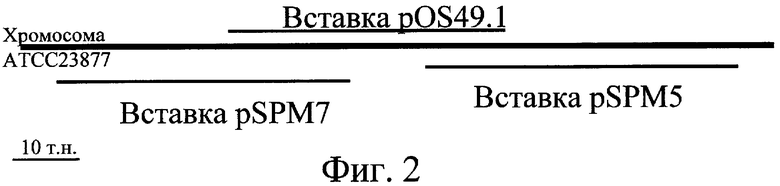
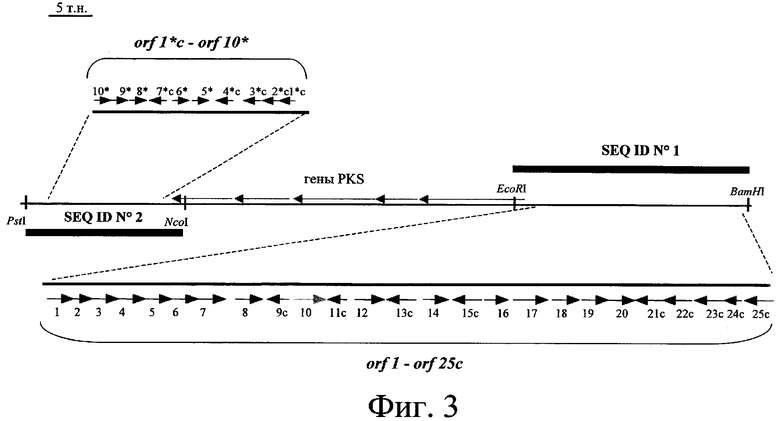
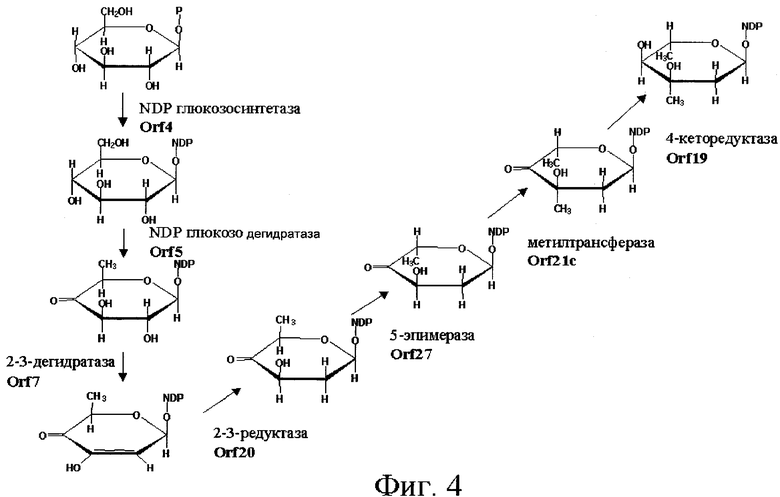
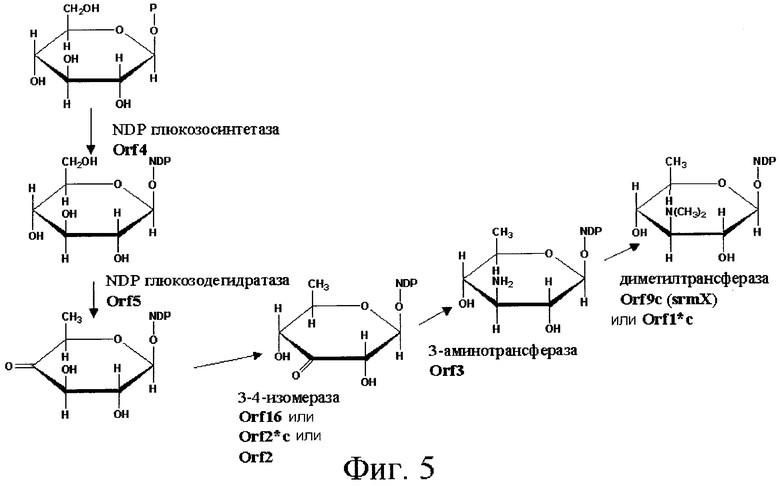
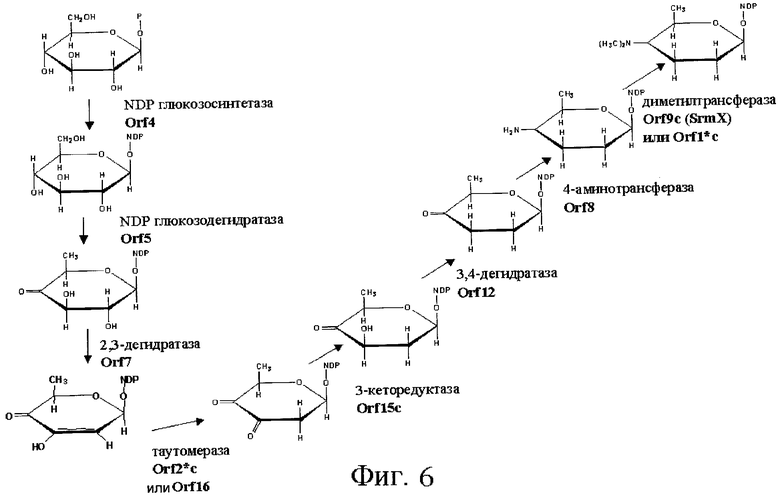
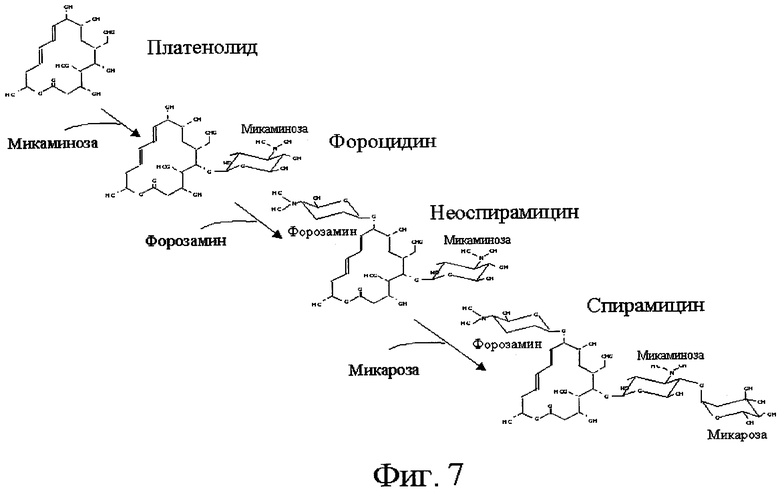
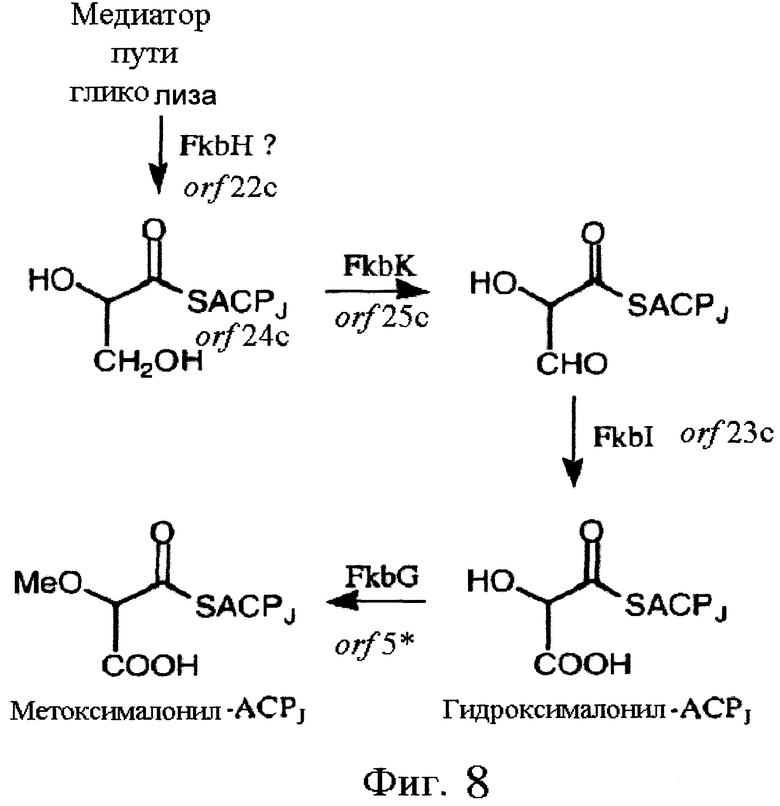
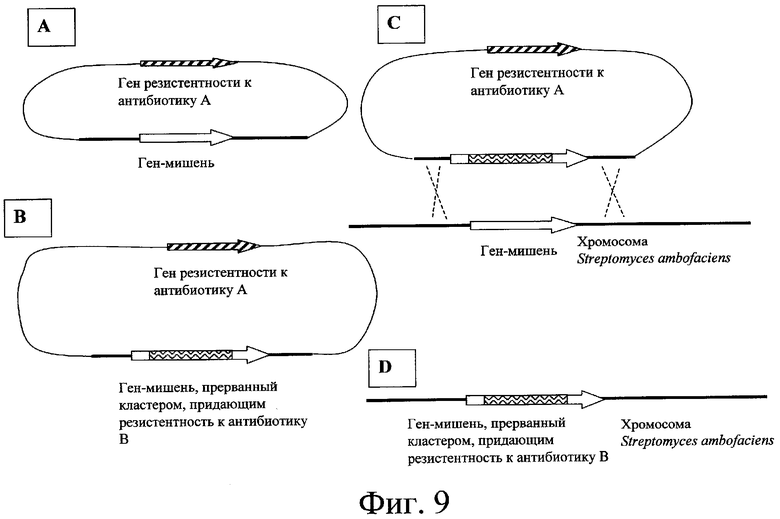
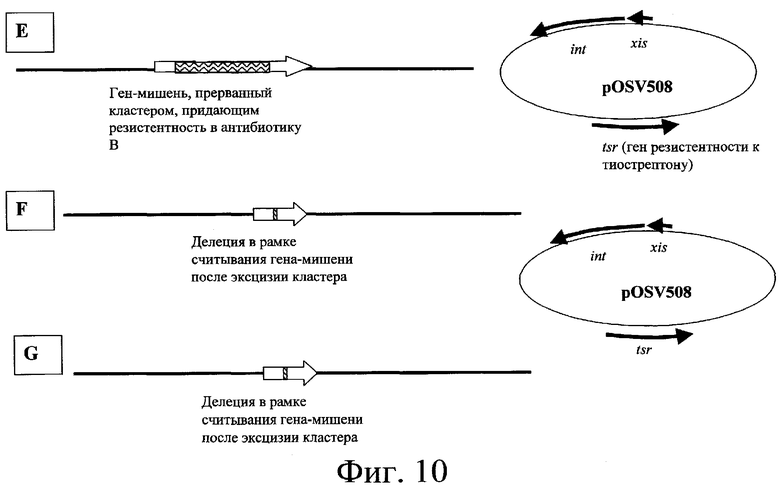
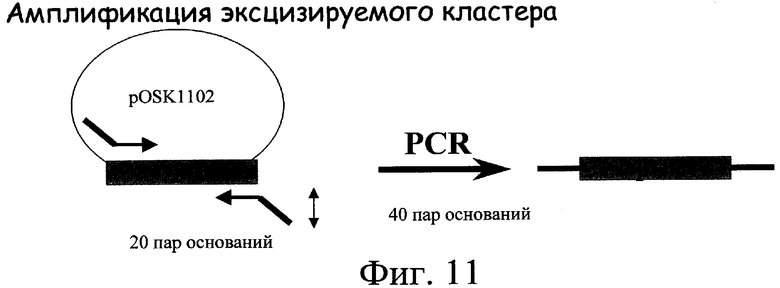
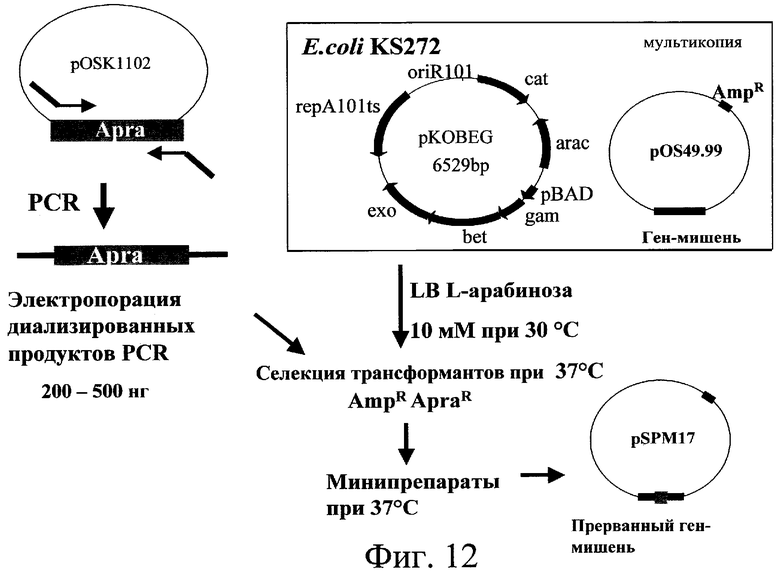
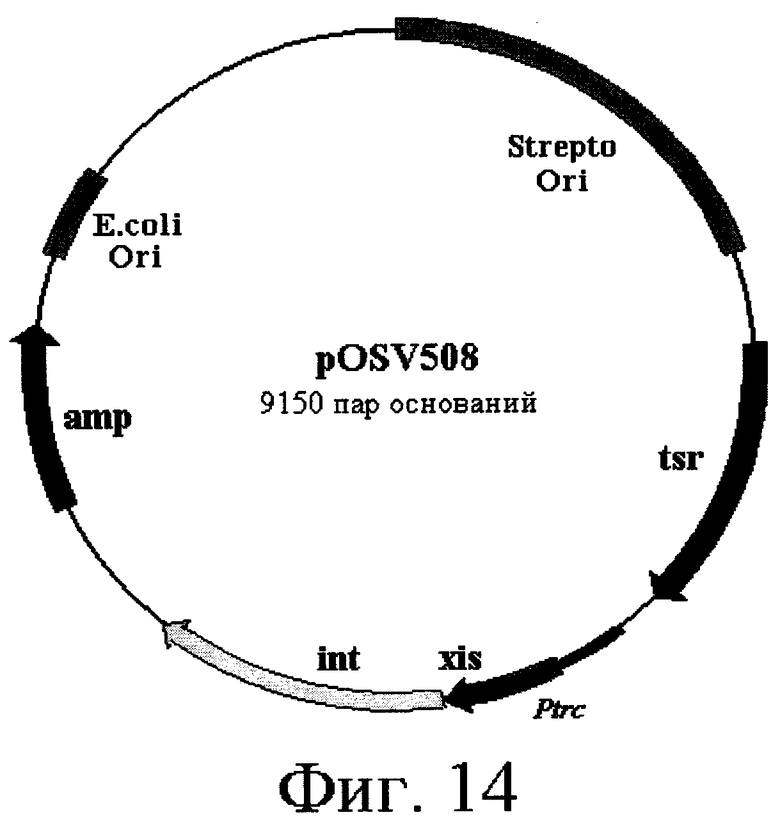
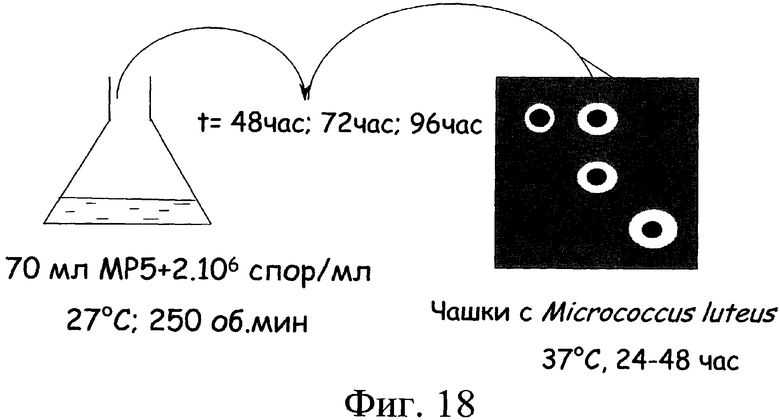
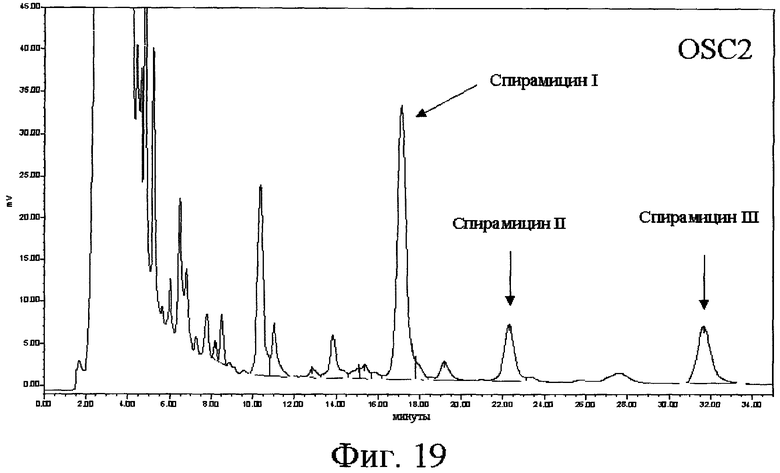
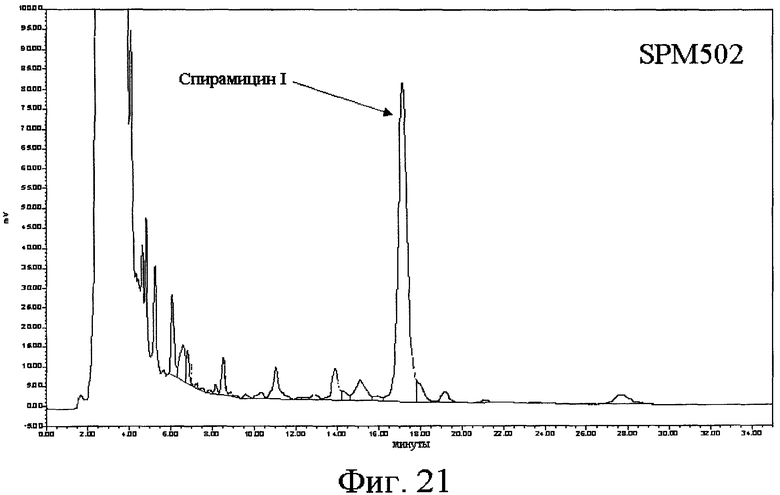
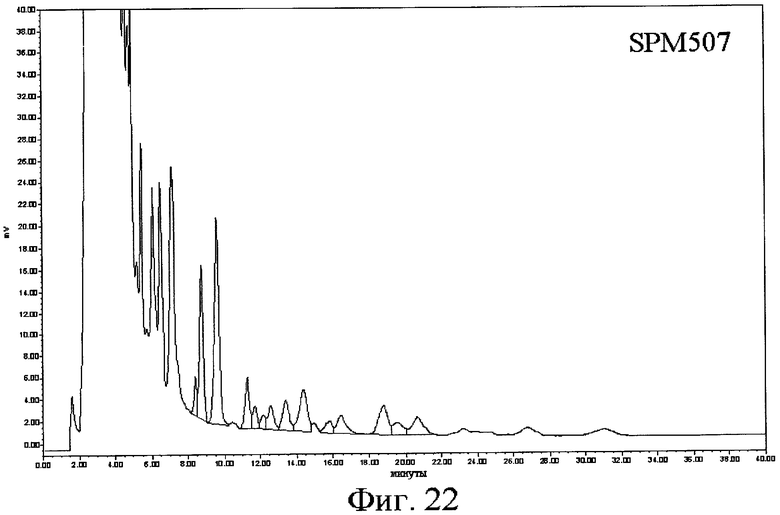
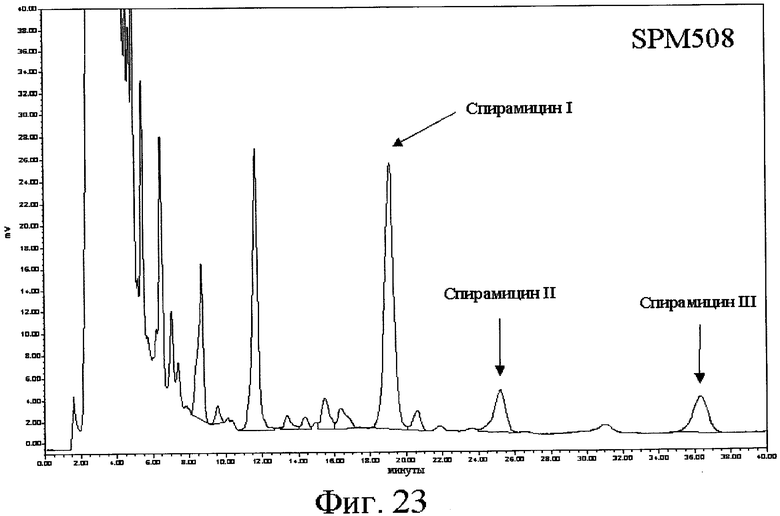
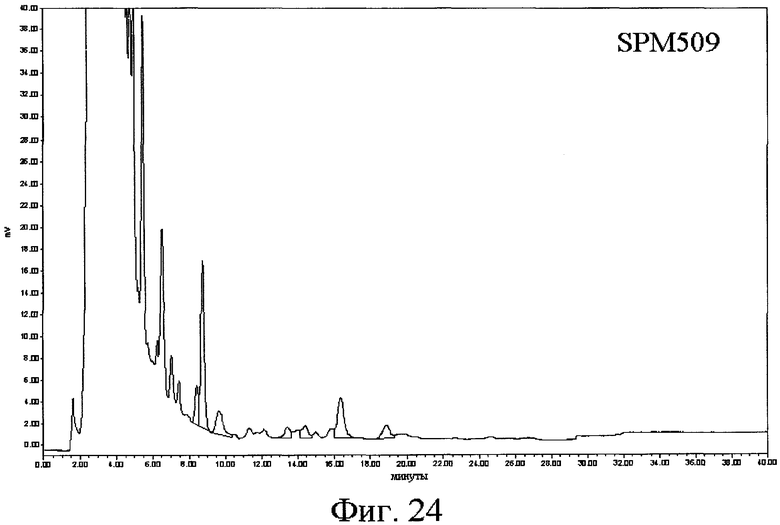
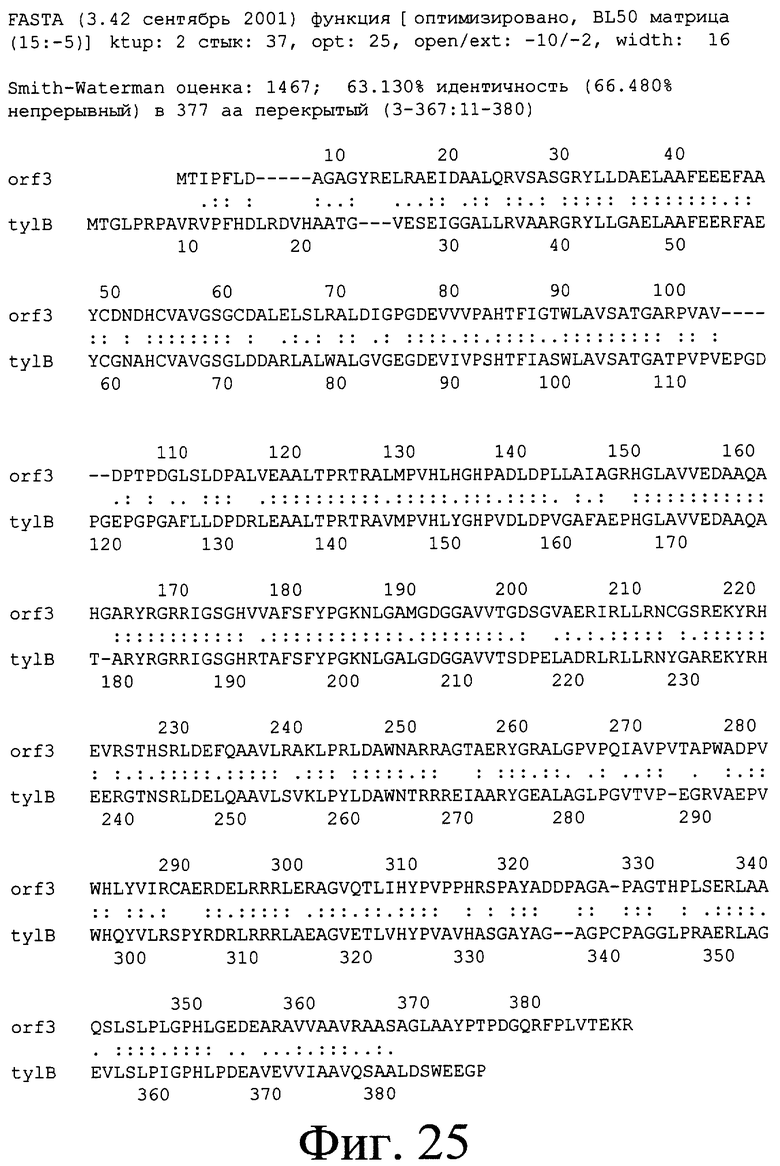

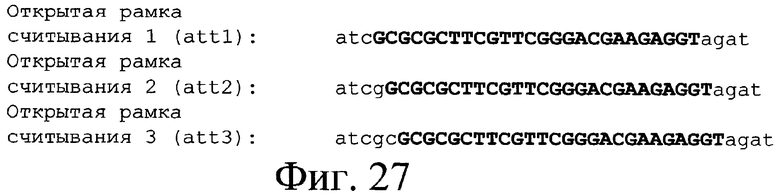
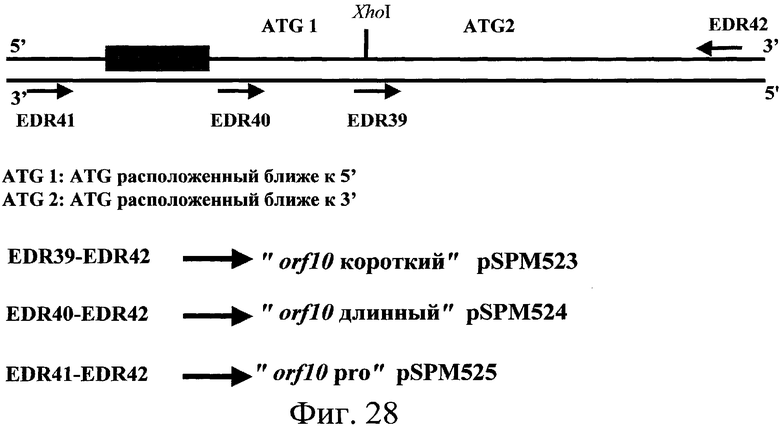
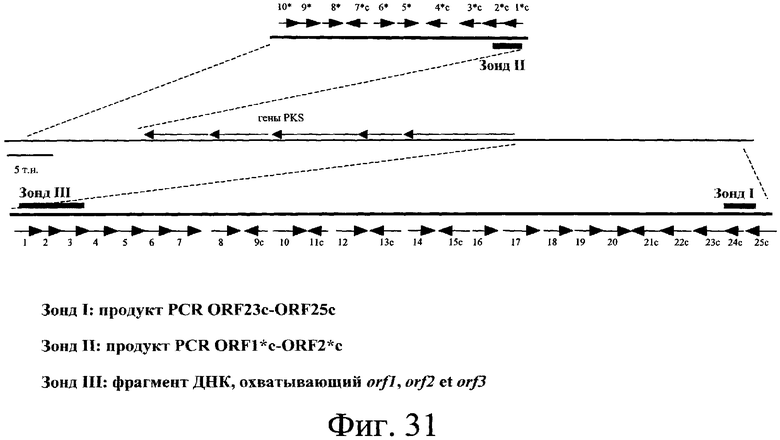

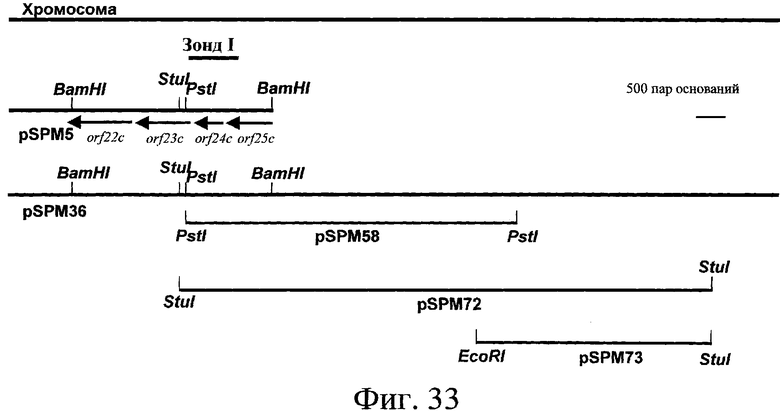
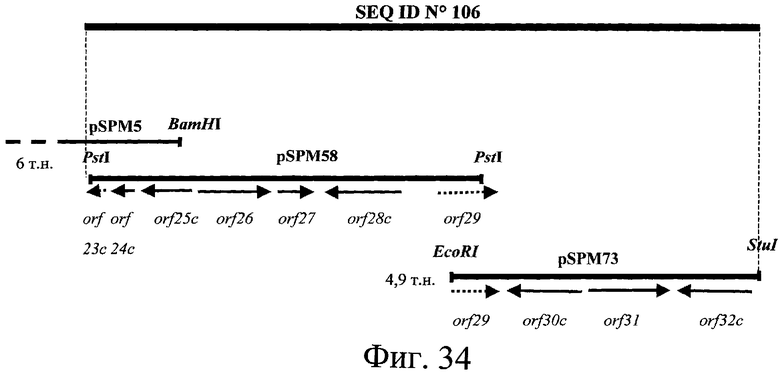
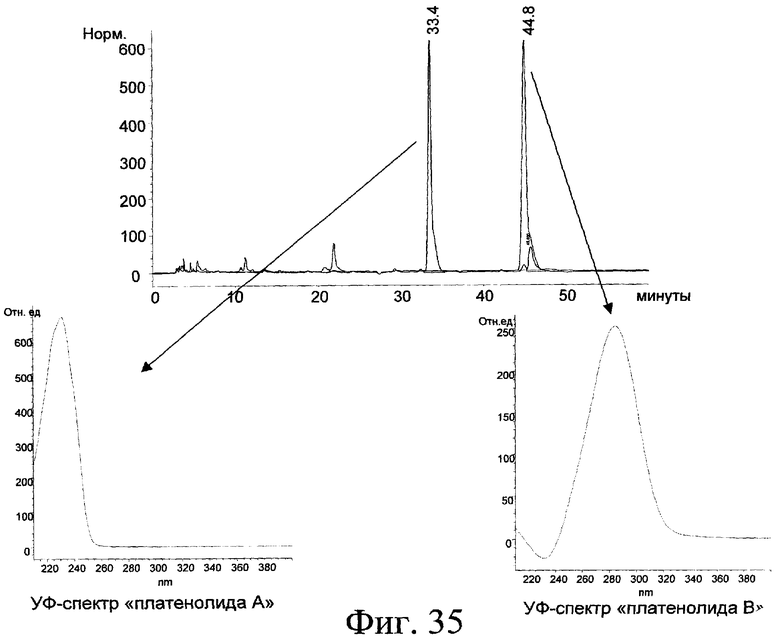
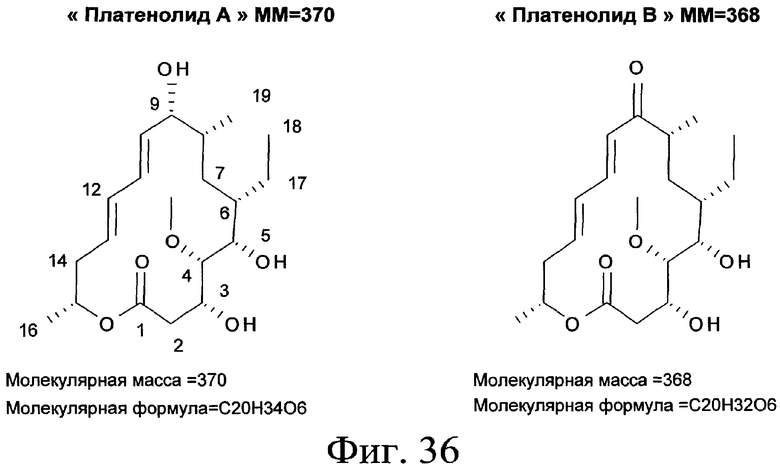
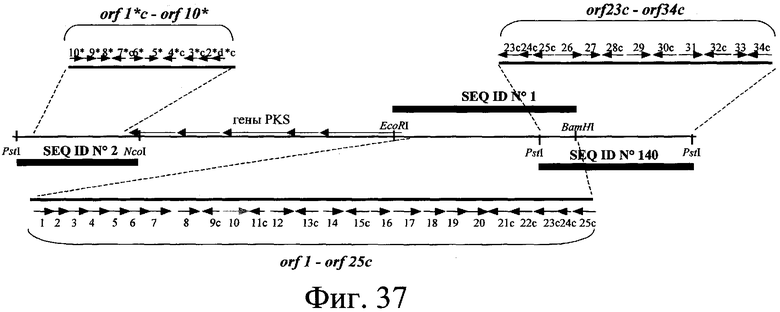
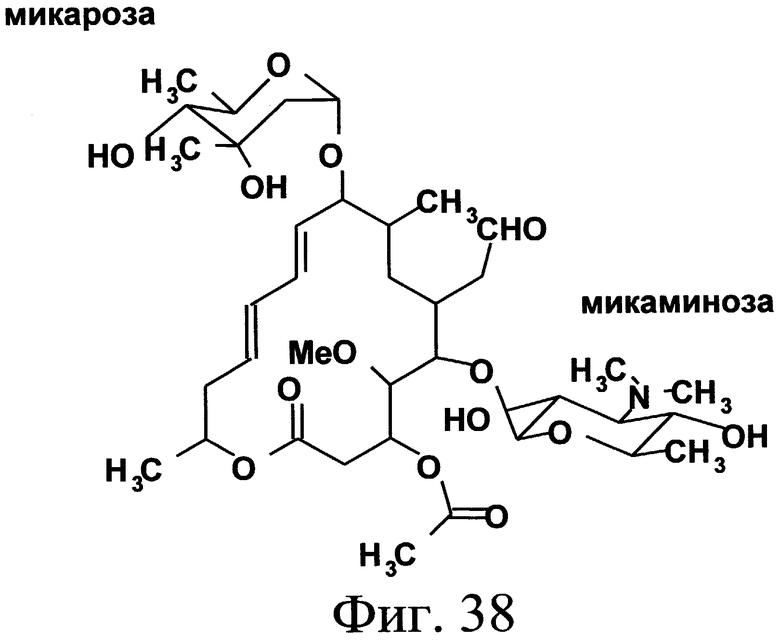
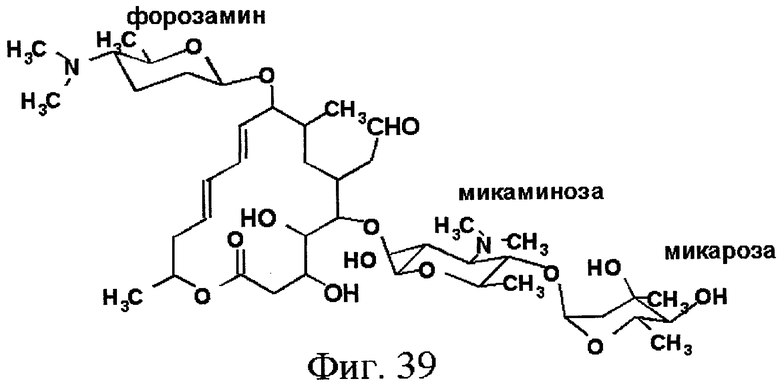
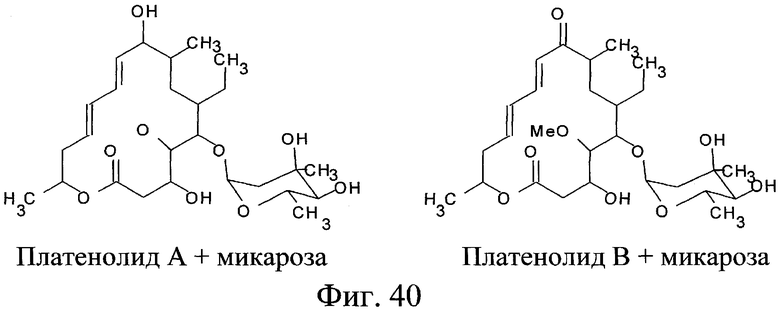
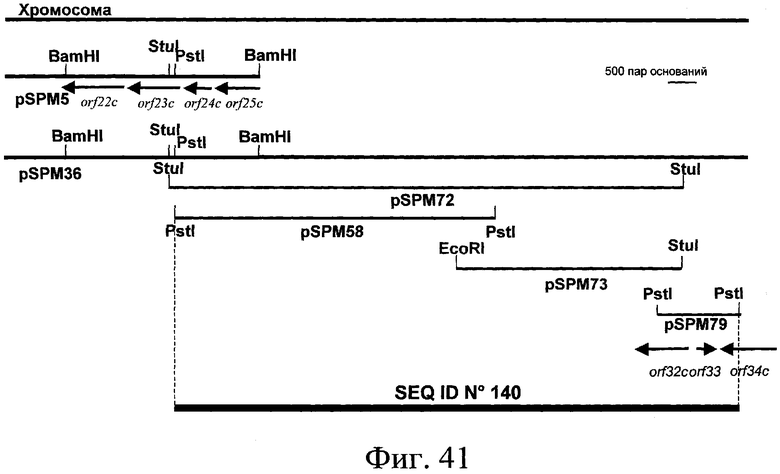
Изобретение относится к области биотехнологии, конкретно к выделению и идентификации новых генов пути биосинтеза спирамицинов и к новым полипептидам, участвующим в этом биосинтезе, и может быть использовано для получения ацилтрансферазы, отвечающей за модификацию платенолида в положении 3. Полинуклеотидом, кодирующим ацилтрансферазу, которая отвечает за модификацию платенолида в положении 3, трансформируют клетки бактерии рода Streptomyces и получают штамм-продуцент целевого полипептида. Изобретение позволяет повысить степень продуцирования и чистоту продуцируемого спирамицина. 18 н. и 8 з.п. ф-лы, 41 ил., 44 табл.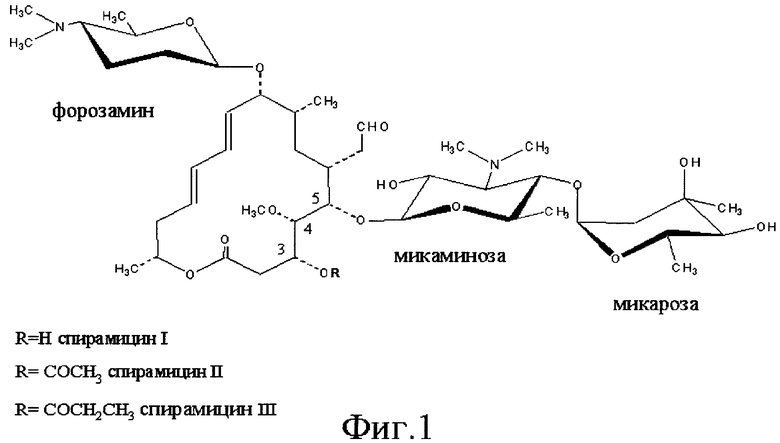
1. Полинуклеотид, кодирующий ацилтрансферазу, отвечающую за
модификацию платенолида в положении 3, причем последовательность вышеуказанного полинуклеотида представляет собой:
(a) одну из последовательностей SEQ ID №111 или 141;
(b) одну из последовательностей, являющихся производными последовательностей (а) в соответствии с вырожденностью генетического кода.
2. Полинуклеотид, кодирующий ацилтрансферазу, отвечающую за модификацию платенолида в положении 3, гибридизующийся в условиях высокой жесткости по меньшей мере с одним из полинуклеотидов по п.1, причем условия высокой жесткости подразумевают осуществление гибридизации при температуре от 55 до 65°С с последующими одной или несколькими промывками, осуществляемыми в буфере 2Х SSC при температуре от 55 до 65°С, затем с одной или несколькими промывками, осуществляемыми в буфере 0,5Х SSC при температуре от 55 до 65°С.
3. Полинуклеотид, обладающий по меньшей мере 70, 80, 85, 90, 95 или 98% идентичности по нуклеотидам с полинуклеотидом по п.1 и кодирующий ацилтрансферазу, отвечающую за модификацию платенолида в положении 3.
4. Полинуклеотид по п.2 или 3, отличающийся тем, что он выделен из бактерии рода Streptomyces.
5. Полинуклеотид по пп.2, 3 или 4, отличающийся тем, что он кодирует протеин, участвующий в биосинтезе макролида.
6. Полинуклеотид по пп.2, 3, 4 или 5, отличающийся тем, что он кодирует протеин, обладающий активностью, подобной протеину, кодируемому полинуклеотидом, с которым он гибридизуется или обладает идентичностью.
7. Полипептид, образующийся в результате экспрессии полинуклеотида по п.1 и представляющий собой ацилтрансферазу, отвечающую за модификацию платенолида в положении 3.
8. Полипептид, представляющий собой ацилтрансферазу, отвечающую за модификацию платенолида в положении 3, причем последовательность вышеуказанного полипептида представляет собой:
(a) одну из последовательностей SEQ ID №112 или 142;
(b) одну из последовательностей, таких, как указанные в (а), за исключением того, что по всей длине вышеуказанной последовательности одна или несколько аминокислот заменены, встроены или подвергнуты делеции без затрагивания функциональных свойств.
9. Полипептид, представляющий собой ацилтрансферазу, отвечающую за модификацию платенолида в положении 3 и обладающий по меньшей мере 70, 80, 85, 90, 95 или 98% идентичности по аминокислотам с полипептидом по п.8.
10. Полипептид по п.9, отличающийся тем, что он выделен из бактерии рода Streptomyces.
11. Полипептид по п.10, отличающийся тем, что он участвует в биосинтезе макролида.
12. Полипептид по п.9, отличающийся тем, что он обладает активностью, подобной активности полипептида, с которым он имеет идентичность.
13. Бактериальный экспрессионный вектор, включающий по меньшей мере одну нуклеотидную последовательность, кодирующую полипептид по п.7 или 8.
14. Бактериальная экспрессионная система, включающая экспрессирующий вектор по п.13 и клетку бактерии рода Streptomyces, позволяющая осуществлять экспрессию одного или нескольких полипептидов по п.7 или 8.
15. Экспрессионная система по п.14, отличающаяся тем, что ее выбирают из прокариотных экспрессионных систем.
16. Бактериальная клетка-хозяин, представляющая собой клетку бактерии рода Streptomyces, для экспрессии полипептида по п.7 или 8, в которую введен по меньшей мере один полинуклеотид и/или по меньшей мере один экспрессирующий вектор по п.1.
17. Способ продуцирования полипептида по п.7 или 8, включающий следующие стадии:
a) инсерция полинуклеотида по п.1 в экспрессирующий вектор;
b) культивирование, на подходящей для экспрессии культуральной среде, клетки бактерии рода Streptomyces, предварительно трансформированной или трансфицированной вектором со стадии а);
c) рекуперация кондиционированной культуральной среды или клеточного экстракта;
d) выделение и очистка из вышеуказанной культуральной среды или из клеточного экстракта, полученной(ного) на стадии с), вышеуказанного полипептида.
18. Штамм Streptomyces ambofaciens, суперэкспрессирующий спирамицины, представляющий собой штамм, депонированный в Национальной коллекции культур микроорганизмов (CNCM) в Институте Пастера, 25 рю дю Доктэр Ру 75724 Париж Cedex 15, Франция, 6 октября 2003 г под регистрационным номером I-3101.
19. Применение полинуклеотида по п.1 для повышения продуцирования макролидов в клетках бактерии рода Streptomyces.
20. Применение последовательности по п.1 в рекомбинантной ДИК или в векторе для продуцирования спирамицинов I, II и III.
21. Полинуклеотид, кодирующий ацилтрансферазу, отвечающую за модификацию платенолида в положении 3, представляющий собой полинуклеотид, комплементарный полинуклеотиду по п.1.
22. Клетка бактерии Streptomyces ambofaciens, являющаяся продуцентом по меньшей мере одного спирамицина, суперэкспрессирующая:
ген, который может быть получен за счет амплификации путем полимеразной цепной реакции (PCR) при использовании пары праймеров, имеющих следующие последовательности:
5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID №138) и
5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID №139),
и в качестве матрицы космиды pSPM36 или полной ДНК Streptomyces ambofaciens;
или ген, являющийся его производным в соответствии с вырожденностью генетического кода.
23. Клетка по п.22, отличающаяся тем, что суперэкспрессии вышеуказанного гена достигают путем трансформации вышеуказанного микроорганизма экспрессирующим вектором.
24. Бактериальный экспрессирующий вектор, включающий:
полинуклеотид, который может быть получен за счет амплификации путем полимеразной цепной реакции при использовании пары праймеров, имеющих следующие последовательности:
5' AAGCTTGTGTGCCCGGTGTACCTGGGGAGC 3' (SEQ ID №138) и
5' GGATCCCGCGACGGACACGACCGCCGCGCA 3' (SEQ ID №139),
и в качестве матрицы космиды рSРМ36 или полной ДНК Streptomyces ambofaciens;
или фрагмент, включающий полинуклеотидную последовательность по п.1.
25. Клетка-хозяин Streptomyces ambofaciens, суперэкспрессирующая спирамицины, в которую введен вектор по п.24.
26. Способ получения полипептида по п.7, включающий следующие стадии:
a) трансформация клетки бактерии рода Streptomyces с помощью по меньшей мере одного экспрессирующего вектора по п.24;
b) культивирование на соответствующей культуральной среде вышеуказанной клетки бактерии рода Streptomyces;
c) рекуперация кондиционированной культуральной среды или клеточного экстракта;
d) выделение и очистка из вышеуказанной культуральной среды или из клеточного экстракта, полученной(ного) на стадии с), вышеуказанного полипептида.
| Способ и устройство для тепловой обработки суспензий по системе г.с.кучеренко | 1978 |
|
SU791655A1 |
| Способ обработки осадков сточных вод | 1979 |
|
SU791656A1 |
| ЕРР J.K | |||
| et al | |||
| Production of a hybrid macrolide antibiotic in Streptomyces ambofaciens and Streptomyces lividans by introduction of a cloned carbomycin biosynthetic gene from Streptomyces thermotolerans | |||
| Gene, 1989, v.85, n.2, p.293-301 | |||
| HARA O | |||
| et al., A macrolide 3-O-acyltransferase gene from | |||
Авторы
Даты
2009-12-27—Публикация
2003-10-08—Подача