Изобретение относится к компьютерному способу, использующему биохимические базы данных при разработке новых белковых соединений для фармацевтики, биотехнологии и других областей промышленности, а также для научных исследований в медицине, биохимии, молекулярной биологии и генетике, для которых существенно использование новых белковых соединений на основе аминокислот.
Белки, основные строительные и функциональные элементы биосистем, имеют многоуровневую иерархическую структуру. Последовательность аминокислот в белковой цепи определяет первичную структуру белка, порядок сворачивания первичной структуры аминокислот в α-спиральные или β-структурые фрагменты определяет его вторичную структуру, а пространственная укладка α-спиральных или β-структурных фрагментов относительно друг друга в пределах субъединицы - третичную структуру белка. О функциональных свойствах белка судят на основании его третичной структуры. В настоящее время для этих целей путем многоступенчатых процедур производят выделение из биосистем индивидуальных нативных, т.е. сохраняющих свою пространственную конформацию, молекул белка, получают их в кристаллическом виде и проводят исследование кристаллов методом рентгеноструктурного анализа (метод РСА) (Попов Е.М., Демин В.В., Шибанова Е.Д, Проблема белка. Том 2. Пространственное строение белка, М.: Наука, 1996, 480 с.). Полученную информацию о дифракционной картине кристаллов записывают на жесткий носитель в компьютер, и, с помощью специально разработанных программ, производят расшифровку его третичной структуры. На основании полученной структуры, записанной в виде координат его атомов в файлах Protein Data Bank, с помощью специальных компьютерных программ, использующих эти файлы, судят о молекулярных механизмах функционирования того или иного белка. В частности, с использованием расшифрованных третичных структур производят разработку новых лекарственных средств. Однако исследования третичной структуры занимают много времени и являются очень дорогостоящими.
Известен метод секвенирования для определения нуклеотидных последовательностей в ДНК целых геномов (Киселев Л.Л. Геном человека и биология XXI века. - Вестн. Рос. Акад. Наук, т.70, №5, с.412-424). Метод позволяет путем перевода этих последовательностей в цепи аминокислот получать информацию о первичных структурах большого числа новых белков, пространственная структура которых и функциональные свойства остаются неизвестны. При этом число исследований кристаллических белков, далеко не всегда возможное, растет в арифметической прогрессии, а количество известных первичных структур с неизвестной функцией - в геометрической прогрессии. По этой причине актуальной является проблема прогнозирования вторичной структуры белков, т.е. положения α-спиральных или β-структурных фрагментов на основе первичной структуры белков (Финкельштейн А.В., Птицын О.Б. Физика белка. - М.: Книжный дом «Университет», 2002, 376 с.).
Прогнозирование вторичной структуры белка на основе его первичной структуры является одним из необходимых этапов к прогнозированию третичной структуры и функциональных свойств новых белков. Создание точных способов прогнозирования вторичной структуры белка приводит к существенному удешевлению исследований по выяснению их функциональных свойств. Кроме того, использование принципов прогнозирования вторичных структур позволит конструировать такие первичные структуры белков, которые будут обладать заранее заданной вторичной структурой и свойствами. Решение этой проблемы особенно важно в технологии изготовления фармацевтических и иммунологических препаратов белкового происхождения. В частности, иммунные белки можно будет создавать в считанные дни, не прибегая к использованию для этих целей животных, что особенно актуально в периоды эпидемий (например, гриппа).
Известен экспериментальный способ обнаружения α-спиральных или β-структурных фрагментов в белке на установке ядерного магнитного резонанса, предусматривающий измерение значений химических сдвигов ядер атомов в молекуле белка, по которым судят о наличии и расположении α-спиральных или β-структурых участков в его структуре. (Заявка WO 2004011909, «Phase-sensetively detected reduced dimensionality nuclear magnetic resonance spectroscopy for rapid chemical shift assignment and secondary structure determination of proteins», МПК G01R 33/46; G01R 33/465; G01R 33/44, опубл. 05.02.2004).
Недостатком данного способа является низкая точность, а также исключительная сложность и высокая стоимость его технического осуществления.
Известен способ определения α-спиральных и β-структурных участков в белке, предусматривающий выделение фрагментов первичной структуры белка, состоящих из шести аминокислот при поиске начального участка спирали, а затем из четырех аминокислот, с последующим анализом в них состава аминокислот и вычислением значений потенциалов спирализации, по которым судят о вероятности отнесения вторичной структуры фрагментов к α-спиральному или β-структурному типам (Chou P.Y., Fasman G.D. Prediction of protein conformation. - Biochemistry, 1974, V.13, pp.222-245; Chen H., Gu F., Huang Z.). Известен также улучшенный способ, основанный на том же методе (Improved Chou-Fasman method for protein secondary structure prediction. BMC Bioinformatics, - 2006, V.7, Suppl.4, S14). Для определения положения изгибов β-структуры (реверсивных поворотов), определяющих участки, по которым происходит складывание первичных структур в β-структуру, Чоу и Фасман используют метод Льюиса (Lewis P.N., Momany F.A., Scheraga Н.А. Folding of polypeptide chains in proteins: A proposed mechanism for folding. - Proc. Natl. Acad. Sci. USA, 1971, V.68, P.2293).
Однако существующие методы прогнозирования вторичной структуры белка на основе его первичной структуры обладают такими недостатками, как предсказание ложных фрагментов вторичной структуры или неполное предсказание всех фрагментов вторичной структуры, что связано с методологическими недостатками этих подходов (в частности, с вероятностным характером проводимых вычислений).
Задачей предлагаемого изобретения является создание способа прогнозирования вторичной структуры белка, позволяющего получить технический результат, заключающийся в повышении точности прогнозирования вторичной структуры белка, что открывает также путь к конструированию первичных структур белков (дизайну белковых молекул), принимающих в физиологических условиях заданную вторичную структуру.
Способ прогнозирования вторичной структуры белка на основе определения положения α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре белка заключается в следующем:
A) создают базу данных аминокислотных пентафрагментов белков, содержащую папки с пентафрагментами, причем исходный список папок составлен по их названиям, сформированным на основании закодированного в двоичной системе описания водородных связей пептидных групп пентафрагментов во вторичной структуре белков, и записывают ее на информационный носитель;
Б) вводят в память компьютера записанную на информационный носитель базу данных аминокислотных пентафрагментов белков;
B) вводят в память компьютера программу FILEMAKER для представления информации о первичной структуре исследуемого белка в виде рабочего файла;
Г) вводят в память компьютера программу PREDICTOR для выделения пентафрагментов в рабочем файле исследуемого белка, поиска выделенных пентафрагментов в базе данных и записи названий папок базы данных, в которых обнаружены искомые пентафрагменты;
Д) вводят в память компьютера текстовый файл в виде:
- либо последовательности нуклеотидов, кодирующих исследуемый белок или его фрагмент;
- либо последовательности аминокислот исследуемого белка или его фрагмента;
Е) текстовый файл представляют в виде рабочего файла, содержащего последовательность аминокислот исследуемого белка или его фрагмента, с помощью ранее записанной в память компьютера программы FILEMAKER;
Ж) проводят поиск пентафрагментов исследуемого белка в базе данных с помощью ранее записанной в память компьютера программы PREDICTOR, при этом алгоритм программы включает в себя два этапа:
I) проведение поиска начального пентафрагмента, включающее:
- выделение в последовательности аминокислот исследуемого белка первого пентафрагмента;
- запоминание и кодирование этого пентафрагмента с целью проведения поиска в базе данных;
- проведение поиска первого пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы;
- при нахождении первого пентафрагмента в базе данных на основе исходного списка папок считают этот фрагмент начальным и производят:
- фиксирование номера папки базы данных, содержащей начальный пентафрагмент;
- внесение номера папки базы данных, содержащей начальный пентафрагмент, в рабочий файл исследуемого белка;
- при не нахождении первого пентафрагмента в базе данных на основе исходного списка папок производят:
- сдвиг вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение следующего по порядку пентафрагмента;
- запоминание и кодирование этого пентафрагмента с целью проведения поиска в базе данных;
- проведение поиска следующего пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы;
- повторение поиска начального пентафрагмента до нахождения искомого пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы, и назначение найденного пентафрагмента начальным;
II) проведение поиска последующих пентафрагментов после нахождения начального пентафрагмента, включающее:
- при совпадении начального пентафрагмента с первым пентафрагментом в последовательности аминокислот исследуемого белка производят:
- сдвиг вперед вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
- запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
- создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
- проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
- фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска последующих пентафрагментов до конца последовательности аминокислот исследуемого белка;
- при несовпадении начального пентафрагмента с первым пентафрагментом в последовательности аминокислот исследуемого белка производят:
- сдвиг вперед вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
- запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
- создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
- проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
- фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска последующих пентафрагментов до конца последовательности аминокислот исследуемого белка;
- возврат к найденному начальному пентафрагменту;
- сдвиг назад вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
- запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
- создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
- проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
- фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска пентафрагмента до начала последовательности аминокислот исследуемого белка;
З) прогнозируют вторичную структуру белка по положению α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре белка, определенному на основе сведений о номерах папок, последовательно внесенных в рабочий файл для всех пентафрагментов исследуемого белка или его фрагмента.
Способ осуществляют следующим образом:
А) создают базу данных аминокислотных пентафрагментов белков, содержащую папки с пентафрагментами, причем исходный список папок составлен по их названиям, сформированным на основании закодированного в двоичной системе описания водородных связей пептидных групп пентафрагментов во вторичной структуре белков, и записывают ее на информационный носитель.
а) из Protein Data Bank производят скачивание находящихся в открытом доступе файлов с координатами атомов кристаллов белков, исследованных методом РСА. Для создания начальной базы было произведено скачивание 500 файлов белков;
б) с помощью компьютерной программы Protein 3D (Компьютерная программа «Protein 3D», Зарегистрировано в Рос АПО, No. 980143 от 03.05.98, авторы: Карасев В.А., Демченко Е.Л.) на основе полученных из Protein Data Bank файлов создают текстовые файлы, содержащие первичные структуры белков с описанием водородных связей, образуемых пептидными группами основных цепей белков во вторичной структуре;
в) с помощью комплекса программ для создания базы проводят следующие действия:
- производят нарезку полученных первичных структур белков на фрагменты из пяти аминокислот (пентафрагменты) таким образом, чтобы каждый последующий фрагмент выделялся со сдвигом на одну аминокислоту по отношению к предыдущему фрагменту, а информация о водородных связях каждого выделяемого фрагмента во вторичной структуре белка полностью сохранялась;
- пентафрагменты, гомологичные по структуре водородных связей пептидных групп во вторичной структуре белка, сортируют на папки, присваивая названиям папок закодированное в двоичной системе описание водородных связей пептидных групп. Наличие водородной связи обозначали цифрой «1», отсутствие водородной связи - цифрой «0».
В каждом пентафрагменте имеется 5 пар пептидных групп, водородные связи которых описываются четырьмя видами пар переменных: 00, 01, 10 и 11. Таким образом, название папки, содержащей гомологичные по структуре пентафрагменты, формируется из 10 символов. Например, номер 1111111111 (для облегчения восприятия мы вводим два интервала - 11 111111 11) соответствует максимальному числу водородных связей пентафрагмента во вторичной структуре белка (в ядре α-спирали), номер 00 000000 00 - отсутствию водородных связей у пентафрагмента в ближайшем окружении основной цепи (в β-структуре), а номер 01 000000 10 - образованию водородной связи в области изгиба β-структуры.
г) производят упрощение выделенных пентафрагментов путем удаления из них информации о структуре водородных связей и оставления только последовательности из пяти аминокислот;
д) с целью облегчения дальнейшей процедуры поиска пентафрагментов в базе данных производят их сортировку на файлы, содержащие фрагменты с одинаковым пятизначным числовым индексом, который им присваивают путем отнесения каждой из аминокислот пентафрагмента к одной из четырех групп преобразований антисимметрии. При этом в имени файла записывают этот пятизначный индекс и название папки, в которой этот файл расположен.
Созданная база данных содержит более 100 тысяч пентафрагментов, сортированных на более чем 500 папок. База данных организована в систему, состоящую из 16 гиперкубов, изоморфных булевым гиперкубам В6.
База данных может постоянно пополняться путем обработки новых файлов из Protein Data Bank. Также может быть создана теоретическая база данных.
Б) вводят в память компьютера записанную на информационный носитель базу данных аминокислотных пентафрагментов белков;
В) вводят в память компьютера программу FILEMAKER для представления информации о первичной структуре исследуемого белка в виде рабочего файла;
Компьютерная программа FILEMAKER является вспомогательной и предназначена для представления информации о первичной структуре белка, записанной в разных форматах в файлах банков данных (например, в Genbank), в виде рабочих файлов, формат которых пригоден для использования программой PREDICTOR. Программа FILEMAKER может использовать текстовые файлы, содержащие либо последовательность нуклеотидов, кодирующих исследуемый белок или его фрагмент, либо последовательность аминокислот исследуемого белка или его фрагмента;
Г) вводят в память компьютера программу PREDICTOR для выделения пентафрагментов в рабочем файле исследуемого белка, поиска выделенных пентафрагментов в базе данных и записи названий папок базы данных, в которых обнаружены искомые пентафрагменты;
Компьютерная программа PREDICTOR написана на основе алгоритма, применяемого в предлагаемом способе прогнозирования вторичной структуры белка, использует для своей работы формат файлов базы данных пентафрагментов белков и формат рабочих файлов, созданных программой FILEMAKER.
Программа проводит следующие операции:
- в последовательности аминокислот исследуемого белка выделяет начальный пентафрагмент;
- запоминает выделенный пентафрагмент для целей поиска в базе данных;
- кодирует этот пентафрагмент для целей поиска в базе данных;
- проводит поиск пентафрагмента в базе данных на основе исходного списка папок, введенного в текст программы;
- фиксирует номер папки, содержащей найденный пентафрагмент;
- записывает номер папки, содержащей найденный пентафрагмент, в рабочий файл;
- осуществляет сдвиг вдоль последовательности аминокислот исследуемого белка на одну аминокислоту в рабочем файле, и выделяет в последовательности аминокислот следующий пентафрагмент;
- запоминает и кодирует новый пентафрагмент с целью проведения его поиска в базе данных;
- на основе номера папки, содержащей найденный пентафрагмент, создает новый список папок для поиска следующего пентафрагмента и повторяет всю процедуру;
- проводит поиск каждого выделенного нового пентафрагмента до конца последовательности аминокислот исследуемого белка.
Д) вводят в память компьютера текстовый файл в виде:
- либо последовательности нуклеотидов, кодирующих исследуемый белок или его фрагмент;
- либо последовательности аминокислот исследуемого белка или его фрагмента;
Тестовый файл представляет собой файл, скачанный из GenBank, GenPept, FASTA, Protein Data Bank из домена http://www.ncbi.nlm.nih.gov или файл из базы данных, созданной исследовательским путем.
Е) текстовый файл представляют в виде рабочего файла, содержащего последовательность аминокислот исследуемого белка или его фрагмента, с помощью ранее записанной в память компьютера программы FILEMAKER;
Формат рабочего файла показан в таблице 1.
Запись последовательности аминокислот исследуемого белка в рабочем файле производится снизу вверх, что отражает порядок синтеза белка на рибосоме (приращение белковой последовательности происходит в процессе биосинтеза со стороны прикрепленного к рибосоме C-конца). Столбцы файла имеют следующую нумерацию:
1 - номера аминокислот в исследуемом белке, записанные снизу вверх;
2 - триплеты, кодирующие последовательность аминокислот исследуемого белка;
3 - последовательность аминокислот, записанная однобуквенными обозначениями;
4 - последовательность аминокислот, записанная трехбуквенными обозначениями;
5 - столбец для записи результатов поиска пентафрагментов, соответствует десятизначным номерам папок базы данных, в которых найдены искомые пентафрагменты.
В нулевой строке находится сигнальное значение начала последовательности (МЕТ), в строке N - сигнальное значение конца белковой последовательности (STP).
Ж) проводят поиск пентафрагментов исследуемого белка в базе данных с помощью ранее записанной в память компьютера программы PREDICTOR, при этом алгоритм программы включает в себя два этапа:
I) проведение поиска начального пентафрагмента, включающее:
- выделение в последовательности аминокислот исследуемого белка первого пентафрагмента;
Программа выделяет первый пентафрагмент (Табл. 1, номера с 1 по 5). Нулевая строка, означающая начало последовательности аминокислот, в рабочем файле и не читается.
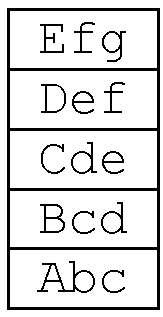
- запоминание и кодирование этого пентафрагмента с целью проведения поиска в базе данных;
Программа запоминает первый пентафрагмент сверху вниз и записывает в память компьютера в последовательности слева направо: Efg, Def, Cde, Bcd, Abc.
Процедура кодирования пентафрагмента для поиска в базе данных связана с особенностью обозначения файлов с пентафрагментами в базе данных, аналогична порядку обозначения пентафрагментов в базе данных (пункт А), подпункт д):
- каждой аминокислоте пентафрагмента сверху вниз программа присваивает номер группы преобразований антисимметрии, в которую она входит (Карасев В.А., Лучинин В.В. Введение в конструирование бионических наносистем. - М.: Физматлит, 2009, 464 с.);
- номер, записанный слева направо, используется программой для поиска номера файла, содержащего искомый пентафрагмент, в папках базы данных.
- проведение поиска первого пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы;
Поиск первого пентафрагмента в папках базы данных на основе исходного списка папок состоит в следующем:
В текст программы введен исходный список папок, который включает две группы:
1 группа - 00 000000 00, 01 000000 00, 10 000000 00, 11 000000 00;
2 группа - 11 111111 11,10 111111 11, 01 111111 11, 00 111111 11.
Выбор этих двух групп обусловлен наличием в белках двух наиболее распространенных типов вторичных структур - β-структур (папка с пентафрагментами 00 000000 00) и α-спиралей (папка с пентафрагментаи 11 111111 11), а также ближайших к ним модификаций (для 00 000000 00 это папки 01 000000 00, 10 000000 00 и 11 000000 00, а для 11 111111 11 - папки 10 111111 11, 01 111111 11 и 00 111111 11).
Программа просматривает последовательно содержимое базы данных на основе исходного списка папок и сверяет пятизначный номер, присвоенный первому пентафрагменту с пятизначным номером файла в означенных папках базы данных. При нахождении файлов с пятизначным номером в одной или нескольких папках списка программа сверяет запомненную последовательность аминокислот в первом пентафрагменте с последовательностями аминокислот пентафрагментов в просматриваемых файлах.
- при нахождении первого пентафрагмента в базе данных на основе исходного списка папок считают этот фрагмент начальным и производят:
- фиксирование номера папки базы данных, содержащей начальный пентафрагмент;
- внесение номера папки базы данных, содержащей начальный пентафрагмент, в рабочий файл исследуемого белка;
- при не нахождении первого пентафрагмента в базе данных на основе исходного списка папок производят:
- сдвиг вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение следующего по порядку пентафрагмента;
- запоминание и кодирование этого пентафрагмента с целью проведения поиска в базе данных;
- проведение поиска следующего пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы;
- повторение поиска начального пентафрагмента до нахождения искомого пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы, и назначение найденного пентафрагмента начальным;
II) проведение поиска последующих пентафрагментов после нахождения начального пентафрагмента, включающее:
- при совпадении начального пентафрагмента с первым пентафрагментом в последовательности аминокислот исследуемого белка производят:
- сдвиг вперед вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
- запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
- создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
На основе номера папки, в которой был найден пентафрагмент, программа создает новый список из четырех папок путем приписывания к этому номеру с левой стороны пар переменных в последовательности 00, 01, 10 и 11 и удаления одной пары переменных с правой стороны.
- проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
- фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска последующих пентафрагментов до конца последовательности аминокислот исследуемого белка;
- при несовпадении начального пентафрагмента с первым пентафрагментом в последовательности аминокислот исследуемого белка производят:
- сдвиг вперед вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
- запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
- создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
- проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
- фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска последующих пентафрагментов до конца последовательности аминокислот исследуемого белка;
- возврат к найденному начальному пентафрагменту;
Найденный начальный пентафрагмент служит основой для продолжения работы программы сторону начала последовательности аминокислот исследуемого белка, для завершения поиска пентафрагментов в базе данных. Для этого программа производит:
- сдвиг назад вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
- запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
- создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
На основе номера папки, в которой был найден начальный пентафрагмент, программа создает список из четырех папок путем приписывания к этому номеру с правой стороны пар переменных в последовательности 00, 01, 10, 11, и удаления одной пары переменных с левой стороны;
- проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска пентафрагмента до начала последовательности аминокислот исследуемого белка.
З) прогнозируют вторичную структуру белка по положениям α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре белка, определенных на основе сведений о номерах папок, последовательно внесенных в рабочий файл для всех пентафрагментов исследуемого белка или его фрагмента.
В результате действий программы PREDICTOR в рабочем файле оказывается полностью заполненным пятый столбец, на основе которого судят о вторичной структуре анализируемого белка. Так, наличие в столбце идущих подряд папок с нумерацией 00 000000 00 свидетельствует о том, данный фрагмент относится к β-структуре. В то же время, несколько идущих подряд папок с нумерацией 11 111111 11 является основанием к отнесению данного фрагмента к α-спиральному. Ряд папок, которые начинаются с папки 01 000000 00, в середине которых находится папка 10 000000 01, а заканчиваются папкой 00 000000 10, относится к участку формирования изгиба β-структуры. Существуют варианты изгибов β-структуры. Переходные между α-спиральной и β-структурной конформации также описываются соответствующими папками. Более детально этот вопрос рассмотрен в примерах.
Пример 1.
В данном примере рассмотрен способ, иллюстрирующий ситуацию, когда начальный пентафрагмент совпадает с первым фрагментом анализируемого белка.
Проводили прогнозирование положения α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре репрессора метионина (публикация: Rafferty J.B., Somers W.S., Saint-Girons I., Phillips S.E.V. Three dimensional crystal structures of Escherichia coli met repressor with and without corepressor. Nature, 1989, V. 341, p.705). Репрессор метионина состоит из 105 аминокислот, его третичная структура изучена с разрешением 1,8 Å (индекс в Protein Data Bank - lcmb). Может служить для сопоставления с результатами прогнозирования его вторичной структуры программой PREDICTOR.
В соответствии в описанным выше способом прогнозирования вторичной структуры белка на основе определения положения α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре белка производили:
A) создали базу данных аминокислотных пентафрагментов белков, содержащую папки с пентафрагментами, причем исходный список папок составлен по их названиям, сформированным на основании закодированного в двоичной системе описания водородных связей пептидных групп пентафрагментов во вторичной структуре белков, и записывают ее на информационный носитель;
Б) ввели в память компьютера записанную на информационный носитель базу данных аминокислотных пентафрагментов белков;
B) ввели в память компьютера программу FILEMAKER для представления информации о первичной структуре исследуемого белка в виде рабочего файла;
Г) ввели в память компьютера программу PREDICTOR для выделения пентафрагментов в рабочем файле исследуемого белка, поиска выделенных пентафрагментов в базе данных и записи названий папок базы данных, в которых обнаружены искомые пентафрагменты;
Д) ввели в память компьютера текстовый файл в виде последовательности нуклеотидов, кодирующих исследуемый белок:
Для исследуемого белка информация о последовательности нуклеотидов,
кодирующих последовательность аминокислот (GenBank Ген СР00 1665.1), имеет следующий вид:
1 atggctgaat ggagcggcga atatatcagc ccatacgctg agcacggcaa gaagagtgaa
61 caagtcaaaa agattacggt ttccattcct cttaaggtgt taaaaatcct caccgatgaa
121 cgcacgcgtc gtcaggtgaa caacctgcgt cacgctacca acagcgagct gctgtgcgaa
181 gcgtttctgc atgcctttac cgggcaacct ttgccggatg atgccgatct gcgtaaagag
241 cgcagcgacg aaatcccgga agcggcaaaa gagatcatgc gtgagatggg gattaacccg
301 gagacgtggg aatactaa
E) текстовый файл представили в виде рабочего файла, содержащего последовательность аминокислот исследуемого белка с помощью ранее записанной в память компьютера программы FILEMAKER;
В таблице 2 приведены начальный и конечный участки исходного рабочего файла репрессора метионина, полученного программой FILEMAKER на основе файла GenBank: CP001665.1.
В соответствии с таблицей 1 столбцы файла имеют следующую нумерацию:
1 - номера аминокислот в белке репрессоре метионина, записанные снизу вверх;
2 - триплеты, кодирующие последовательность аминокислот данного белка;
3 - последовательность аминокислот, записанная однобуквенными обозначениями;
4 - последовательность аминокислот, записанная трехбуквенными обозначениями;
5 - столбец для записи результатов поиска пентафрагментов, соответствует десятизначным номерам папок базы данных, в которых найдены искомые пентафрагменты.
Ж) провели поиск пентафрагментов исследуемого белка в базе данных с помощью ранее записанной в память компьютера программы PREDICTOR, при этом алгоритм программы включал в себя два этапа:
I) провели поиск начального пентафрагмента, включающий:
- выделение в последовательности аминокислот исследуемого белка первого пентафрагмента;
В таблице 3 приведены первые шесть аминокислот белка репрессора метионина.
- в анализируемой последовательности аминокислот белка репрессора метионина, начиная с N-конца, программа PREDICTOR выделяет первый пентафрагмент, выделенный в таблице 3 жирным шрифтом:
- запоминание и кодирование этого пентафрагмента с целью проведения поиска в базе данных;
Данный фрагмент запоминается программой в последовательности сверху вниз: Gly, Ser, Trp, Glu, Ala.
Каждая из 20 аминокислот, которые присутствуют в белках, входит в свою группу преобразований (Карасев В.А., Лучинин В.В. Введение в конструирование бионических наносистем. - М.: Физматлит, 2009. - 464 с.):
1 группа - Gly, Pro;
2 группа - Ala, Leu;
3 группа - Ser, Thr, Cys, Met, His, Trp, Phe, Tyr;
4 группа - Asp, Glu, Asn, Gln, Arg, Lys, Val, Ile.
Программа присваивает каждой аминокислоте пентафрагмента номер той группы преобразований, в которую она входит. Для первого пентафрагмента это:
и записывает его в память компьютера, слева направо путем считывания сверху вниз: 13342.
- проведение поиска первого пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы;
Исходный список папок включает две группы: 1 группа - 00 000000 00, 01 000000 00, 10 000000 00, 11 000000 00; 2 группа - 11 111111 11, 10 111111 11, 01 111111 11, 00 111111 11. В данном примере программа ведет поиск запомненного кодового номера пентафрагмента в папках данного списка и находит его в первой группе папок, в папке 00 000000 00. В этой папке имеется ряд файлов, среди которых находится файл с запомненным кодовым номером 13342 (выделен жирным шрифтом):

В файле 13342_0000000000.txt обнаруживается следующая последовательность аминокислот, читаемая сверху вниз:
Gly
Ser
Trp
Glu
Ala
Данная последовательность совпадает с запомненной последовательностью выделенного пентафрагмента: Gly, Ser, Trp, Glu, Ala. Это означает, что искомый первый пентафрагмент найден в папке 00 000000 00.
- при нахождении первого пентафрагмента в базе данных на основе исходного списка папок считают этот фрагмент начальным и производят:
- фиксирование номера папки базы данных, содержащей начальный пентафрагмент;
- внесение номера папки базы данных, содержащей начальный пентафрагмент, в рабочий файл исследуемого белка;
В таблице 4 приведен пример записи начального пентафрагмента (номера папки с найденным пентафрагментом 00 000000 00) в рабочем файле репрессора метионина (см. пятую строку, выделенную жирным шифтом, пятый столбец).
II) провели поиск последующих пентафрагментов после нахождения начального пентафрагмента, включающий:
- при совпадении начального пентафрагмента с первым пентафрагментом в последовательности аминокислот исследуемого белка производят:
- сдвиг вперед вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
В таблице 4 показан новый пентафрагмент, сдвинутый вперед вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделенный программой (строки 2-6, жирный шрифт).
Ниже он показан отдельно:
- запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
Новый пентафрагмент запоминается программой сверху вниз: Glu, Gly, Ser, Trp, Glu, и кодируется (цифры справа). Запоминается его кодовый номер: 41334.
- создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
Папка, содержащая ранее найденный начальный пентафрагмент (он является в данном случае начальным), имеет номер 00 000000 00. На основе этого номера программа создает список из четырех папок путем приписывания к этому номеру с левой стороны пар переменных в последовательности 00, 01, 10 и 11 и удаления одной пары переменных с правой стороны. Список имеет следующий состав: 00 000000 00, 01 000000 00, 10 000000 00, 11 000000 00.
- проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
- фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска последующих пентафрагментов до конца последовательности аминокислот исследуемого белка;
В результате проведения данного поиска программой PREDICTOR весь пятый столбец рабочего файла был заполнен найденными номерами папок (табл.5).
З) провели прогнозирование вторичной структуры белка по положению α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре белка, определенных на основе сведений о номерах папок, последовательно внесенных в рабочий файл для всех пентафрагментов исследуемого белка или его фрагмента.
Вторичная структура белка репрессора метионина, т.е. положение α-спиральных, β-структурных фрагментов, и изгибов β-структуры характеризуется следующими особенностями (табл.6), полученными на основе анализа таблицы 5.
Фрагменты α-спирали, выделенные в таблице 5 жирным шрифтом, как показано в таблице 6, начинаются с папок 11 010101 01, ядро спирали - папки 11 111111 11 и конец спирали - папки 10 101010 11. Вид папок начала и конца спирали, в зависимости от типа спиральных фрагментов, может отличаться от канонического. В репрессоре метионина найдено три типичных спиральных фрагмента (см. табл.5 и 6).
Фрагменты β-структуры (см. табл.6) начинаются с папок 00 101010 10, центральная часть - папки 00 000000 00 и конец β-структуры - папки 01 010101 00. В зависимости от расположения β-структуры (в начале или в конце последовательности аминокислот) они могут не иметь начальных или конечных папок. Вид папок начала и
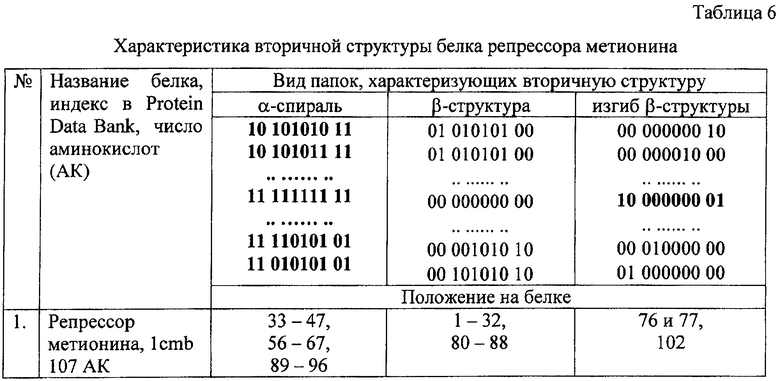
конца β-структуры, также как и в спиралях, может отличаться от канонического. Как видно из таблиц 5 и 6, в репрессоре метионина обнаружено два фрагмента β-структуры - протяженный фрагмент в начале белка и короткий фрагмент - в конце.
Изгиб β-структуры, как показано в таблице 6, начинается с папки 01 000000 00, далее идут папки с перемещением переменной 01 слева направо и центр изгиба - папка 10 000000 01, после чего идут папки с перемещением переменной 10. Заканчивается изгиб папкой 00 000000 10. Могут быть изгибы с повторяющимися подряд двумя и более водородными связями.
В таблице 5 центры изгибов β-структуры выделены жирным шрифтом, они приведены в таблице 6. Изгиб β-структуры в последовательности аминокислот 73-80 с центрами в строках 76 - 10 000001 01, и 77 - 10 100000 01 является примером изгиба с двумя повторяющимися подряд водородными связями. В этом белке имеется также один типичный изгиб с центром в строке 102.
В целом вторичную структуру белка репрессора метионина можно характеризовать как содержащую три α-спиральных, два β-структурных фрагмента и два изгиба β-структуры.
Таким образом, на основе сведений о номерах папок, последовательно внесенных в рабочий файл для всех пентафрагментов, проведено прогнозирование вторичной структуры белка репрессора метионина.
Пример 2.
В данном примере рассмотрен способ, иллюстрирующий ситуацию, когда начальный пентафрагмент не совпадает с первым фрагментом анализируемого белка.
Проводили прогнозирование положения α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре цитохрома C2 Rhodospirillum rubrum (индекс в Protein Data Bank - 2c2c, Salemme F.R., Freer S.T., Xuong N.H., Alden R.A., Kraut J. The structure of oxidized cytochrome с 2 of Rhodospirillum rubrum. J. Biol. Chem. 1971. V.248, P.3910-3921, первичная структура белка: Protein Data Bank - 2C2CA).
В соответствии с описанным выше способом поиска и обнаружения α-спиральных, β-структурых фрагментов и изгибов β-структуры производили следующие процедуры:
Пункты А) - Г) - аналогичны примеру 1.
Д) вводят в память компьютера текстовый файл в виде последовательности аминокислот исследуемого белка: файл из Protein Data Bank - 2C2CA
1 egdaaagekv skkclachtf dqggankvgp nlfgvfenta ahkdnyayse sytemkakgl
61 twteanlaay vknpkafvle ksgdpkaksk mtfkltkdde ienviaylkt lx
E) текстовый файл представляют в виде рабочего файла, содержащего последовательность аминокислот исследуемого белка с помощью ранее записанной в память компьютера программы FILEMAKER;
В таблице 7 приведены начало и конец исходного рабочего файла белка цитохрома C2, полученный программой FILEMAKER на основе файла PDB 2C2CA. Столбцы файла имеют нумерацию согласно таблице 1.
Ж) проводили поиск пентафрагментов исследуемого белка в базе данных с помощью ранее записанной в память компьютера программы PREDICTOR, при этом алгоритм программы включает в себя два этапа:
I) проведение поиска начального пентафрагмента, включающее:
- выделение в последовательности аминокислот исследуемого белка первого пентафрагмента;
В таблице 8 приведены первые шесть аминокислот рассматриваемого белка 2c2c. В анализируемой цепи аминокислот белка 2c2c, начиная с N-конца, программа PREDICTOR выделяет фрагмент первый пентафрагмент:
- запоминание и кодирование этого пентафрагмента с целью проведения поиска в базе данных;
- программа запоминает выделенный пентафрагмент в последовательности сверху вниз: Ala, Ala, Asp, Gly, Glu;
- программа кодирует этот пентафрагмент (номера справа):
и записывает его в память компьютера, слева направо путем считывания сверху вниз: 22414.
- проведение поиска первого пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы;
Исходный список папок:
1 группа - 00 000000 00, 01 000000 00, 10 000000 00, 11 000000 00;
2 группа - 11 111111 11, 10 111111 11, 01 111111 11, 00 111111 11.
В данном примере программа ведет последовательный поиск запомненного кодового номера пентафрагмента в папках данного списка и не находит его ни в первой, ни во второй группе папок.
- при не нахождении первого пентафрагмента в базе данных на основе исходного списка папок производят:
- сдвиг вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение следующего по порядку пентафрагмента;
- запоминание и кодирование этого пентафрагмента с целью проведения поиска в базе данных;
- проведение поиска следующего пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы;
- повторение поиска начального пентафрагмента до нахождения искомого пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы, и назначение найденного пентафрагмента начальным;
В таблице 9 приведен фрагмент рабочего файла белка цитохрома C2, в котором искомый пентафрагмент (выделен жирным шрифтом) был обнаружен в строке 23 (выделена жирным шрифтом). Найденный пентафрагмент назначен в качестве начального.
II) проведение поиска последующих пентафрагментов после нахождения начального пентафрагмента, включающее:
- при несовпадении начального пентафрагмента с первым пентафрагментом в последовательности аминокислот исследуемого белка производят:
- сдвиг вперед вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
- запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
- создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
- проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
- фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска последующих пентафрагментов до конца последовательности аминокислот исследуемого белка;
В таблице 10 приведена процедура поиска пентафрагментов для белка цитохрома C2, от начального пентафрагмента, найденного в 23 строке, до конца последовательности аминокислот исследуемого белка.
- возврат к найденному начальному пентафрагменту;
В таблице 11 приведен этап возврата программы к начальному пентафрагменту, осуществления сдвига назад и выделение в белке нового пентафрагмента. Начальный пентафрагмент (строка 23) выделен жирным шрифтом.
- сдвиг назад вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
Пентафрагмент, выделенный в сторону начала цепи, показан в таблице 11 жирным шрифтом.
- запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
Программа запоминает последовательность: Gln, Asp, Phe, Thr, His.
Кодирование пентафрагмента:
Кодовый номер, запомненный программой: 44333.
- создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
В отличие от процедуры создания нового списка вперед, при перемещении назад список создается путем прибавления к номеру папки начального пентафрагмента, переменных 00, 01, 10, 11 с правой стороны папки, а удаление пары переменных - с левой стороны. Новый список папок для поиска нового пентафрагмента имеет следующий вид: 00 000000 00, 00 000000 01, 00 000000 10,00 000000 11.
- проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
- фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска пентафрагмента до начала последовательности аминокислот исследуемого белка.
В таблице 12 приведен результат поиска пентафрагментов в сторону начала последовательности аминокислот исследуемого белка.
Как следует из приведенной таблицы, процедура нахождения пентафрагментов полностью доходит до первых пяти аминокислот белковой цепи и таким образом процесс анализа рабочего файла считается законченным. Полностью файл результирующий рабочий файл, на основе которого производится прогнозирование вторичной структуры белка цитохрома C2 на основе его первичной структуры, приведен в таблице 13.
З) провели прогнозирование вторичной структуры белка по положению α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре белка, определенных на основе сведений о номерах папок последовательно внесенных в рабочий файл для всех пентафрагментов исследуемого белка или его фрагмента.
Положение α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре цитохрома С2 определено из таблицы 13 и приведено в таблице 14. Из этих данных следует, что в этом белке обнаруживается четыре α-спиральных, два β-структурных фрагмента и два изгиба β-структуры.
111 АК
Пример 3.
В данном примере рассмотрен способ, иллюстрирующий ситуацию, когда проводили прогнозирование положения α-спиральных, β-структурных фрагментов и изгибов β-структурных во фрагменте белка.
В приведенном примере рассмотрен домен (фрагмент) более крупного белка, что показывает возможность использования данного способа не только на целых белках, но и на их фрагментах. Начальный пентафрагмент совпадает с первым фрагментом анализируемого домена.
Проводили прогнозирование положения α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре домена рецептора гормона эстрогена (шифр в Protein Data Bank - 1hcq, публикация SCHWABE J.W.R., CHAPMAN L., FINCH J.T., RHODES D. The crystal structure of the estrogen receptor DNA-binding domain bound to DNA: how receptors discriminate between their response elements. Cell (Cambridge, Mass.), 1993, V.75, P.567).
Фрагменты более крупных белков (домены), часто используют для детального исследования конкретных механизмов взаимодействия рецепторных белков с лигандами. Данный домен, по данным РСА, в кристаллической структуре содержит 74 аминокислоты.
Пункты А) - Г) - аналогичны примеру 1.
Д) вводят в память компьютера текстовый файл в виде последовательности аминокислот фрагмента исследуемого белка: Protein Data Bank - 1HCQA-
1 mketrycavc ndyasgyhyg vwscegckaf fkrsiqghnd ymcpatnqct idknrrkscq
61 acrlrkcyev gmmkggirkd rrgg
Жирным шрифтом выделены аминокислоты, которые в структуре белка отсутствуют. Мы их не использовали.
Е) текстовый файл представляют в виде рабочего файла, содержащего последовательность аминокислот исследуемого белка или его фрагмента, с помощью ранее записанной в память компьютера программы FILEMAKER;
В таблице 15 представлен начальный и конечный участки рабочего файла фрагмента белка рецептора гормона эстрогена (1hcq), полученный на основе последовательности аминокислот из Protein Data Bank.
Ж) проводили поиск пентафрагментов исследуемого белка в базе данных с помощью ранее записанной в память компьютера программы PREDICTOR, при этом алгоритм программы включает в себя два этапа:
I) проведение поиска начального пентафрагмента
В данном примере этот этап аналогичен примеру 1.
II) проведение поиска последующих пентафрагментов после нахождения начального пентафрагмента.
В данном примере этот этап также аналогичен примеру 1.
- повторение поиска последующих пентафрагментов до конца последовательности аминокислот исследуемого белка;
В результате поиска пентафрагментов, проведенного до конца последовательности аминокислот исследуемого фрагмента белка рецептора гормона эстрогена, была получена полностью заполненная таблица 16.
З) прогнозировали вторичную структуру белка по положениям α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре белка, определенных на основе сведений о номерах папок, последовательно внесенных в рабочий файл для всех пентафрагментов исследуемого белка или его фрагмента.
Положение α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре фрагмента белка рецептора гормона эстрогена определено из таблицы 16 и приведено в таблице 17. Из этих данных следует, что в данном белке обнаруживается два α-спиральных, три β-структурных фрагмента и два изгиба β-структуры.
Таким образом, из приведенного примера следует, что прогнозирование вторичной структуры можно проводить и по фрагменту белка.
Сопоставление результатов прогнозирования, полученных предлагаемым способом, с экспериментальными данными
Критерием эффективности предлагаемого способа прогнозирования вторичной структуры белка является сопоставление положения α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре белка, прогнозируемое предлагаемым способом, с данными экспериментального исследования тех же белков, в частности, с помощью метода РСА. Для сравнения мы использовали приведенные выше примеры 1-3 (табл.18).
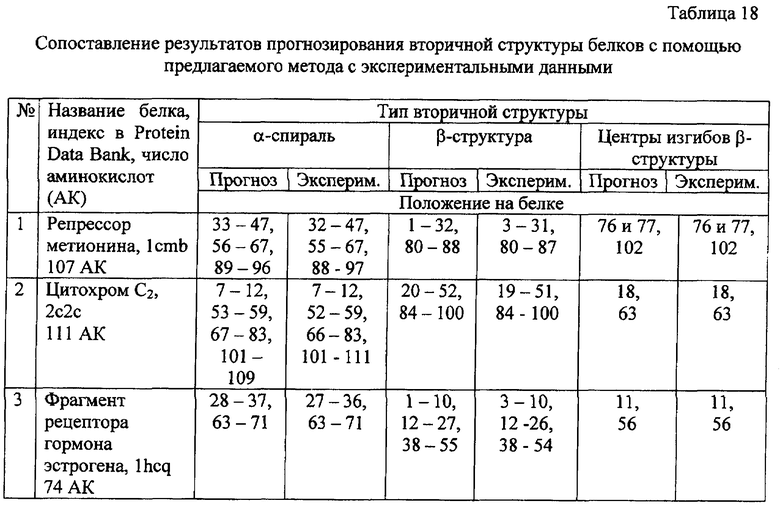
Сопоставление экспериментальных данных и результатов прогнозирования положения α-спиральных, β-структурных фрагментов и изгибов β-структуры с помощью предлагаемого метода показывает, что эти данные практически полностью совпадают (с точностью ± одна аминокислота). Существующие методы прогнозирования вторичной структуры белка на основе его первичной структуры обладают такими недостатками, как предсказание ложных фрагментов вторичной структуры или неполное предсказание всех фрагментов вторичной структуры, что связано с методологическими недостатками этих подходов (в частности, с вероятностным характером проводимых вычислений). При проведении прогнозирования вторичной структуры белка заявляемым способом не обнаруживается ни ложных фрагментов вторичной структуры, ни неполного выявления фрагментов вторичной структуры. Близкая к 100% точность прогнозирования вторичной структуры белка связана с методологическими особенностями данного способа, основанного на выделении идущих со сдвигом в одну аминокислоту пентфрагментов, и их поиске в базе данных пентафрагментов белков, содержащей в названиях папок информацию о водородных связях пентафрагментов во вторичной структуре белка.
То, что результаты прогнозирования положения α-спиральных, β-структурных фрагментов, а также изгибов β-структуры на основе предлагаемого способа обладают высокой точностью и описывают положение водородных связей во вторичной структуре исследуемого белка, создает условия, во-первых, для написания программ, осуществляющих 3D визуализацию прогнозированной структуры, а во-вторых, позволяет использовать эти результаты для последующей разработки способов прогнозирования третичных структур на основе прогнозированных вторичных структур. Тем самым сделан шаг в решении практически важной задачи теоретического построения пространственных структур белков на основе их первичной структуры.
«База данных пентафрагментов белков» и «Компьютерная программа для прогнозирования вторичной структуры белков - PREDICTOR» направлены на регистрацию в Роспатент.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ПРОЕКТИРОВАНИЯ ПЕРВИЧНОЙ СТРУКТУРЫ БЕЛКА С ЗАДАННОЙ ВТОРИЧНОЙ СТРУКТУРОЙ | 2011 |
|
RU2511002C2 |
| АНТИТЕЛО К РЕЦЕПТОРУ ТРОМБОЦИТАРНОГО ФАКТОРА РОСТА (PDGF) И ЕГО ПРИМЕНЕНИЕ | 2019 |
|
RU2778023C1 |
| СПОСОБ ВВЕДЕНИЯ МОЛЕКУЛ GLP-1 | 2003 |
|
RU2332229C2 |
| БИСПЕЦИФИЧЕСКИЕ БЕЛКИ И СПОСОБЫ ИХ ПОЛУЧЕНИЯ | 2017 |
|
RU2745648C2 |
| СПОСОБЫ ЛЕЧЕНИЯ РАССТРОЙСТВ, СВЯЗАННЫХ С FGF21 | 2016 |
|
RU2752530C2 |
| СЛИТЫЙ БЕЛОК, СОДЕРЖАЩИЙ РЕЦЕПТОР TGF-БЕТА, И ЕГО ФАРМАЦЕВТИЧЕСКОЕ ПРИМЕНЕНИЕ | 2018 |
|
RU2776204C1 |
| АНТИТЕЛА-МИМЕТИКИ FGF21 И ПУТИ ИХ ПРИМЕНЕНИЯ | 2018 |
|
RU2774368C2 |
| ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ СЛИТОГО БЕЛКА РЕЦЕПТОРА ТРАНСФОРМИРУЮЩЕГО ФАКТОРА РОСТА БЕТА И ЕЕ ПРИМЕНЕНИЕ | 2019 |
|
RU2791683C2 |
| СПОСОБ ВЫЯВЛЕНИЯ СОЕДИНЕНИЙ, СНИЖАЮЩИХ ФУНКЦИОНАЛЬНУЮ АКТИВНОСТЬ ПРОТЕАЗЫ ВИРУСА ИММУНОДЕФИЦИТА ЧЕЛОВЕКА, И СПОСОБ ИНГИБИРОВАНИЯ ДИМЕРИЗАЦИИ СУБЪЕДИНИЦ ПРОТЕАЗЫ ВИЧ | 2007 |
|
RU2399612C2 |
| БЕЛКИ, СПЕЦИФИЧНЫЕ В ОТНОШЕНИИ CD137 | 2016 |
|
RU2736312C2 |
Изобретение относится к области биоинформатики и биотехнологии, в частности к прогнозированию вторичной структуры белка, и может быть использовано в молекулярной биологии и медицине. Положение α-спиральных, β-структурных фрагментов и изгибов β-структуры в последовательности аминокислот белка прогнозируют с помощью специально написанной программы PREDICTOR путем сравнения выделяемых в рабочем файле исследуемого белка последовательно, со сдвигом в одну аминокислоту фрагментов из пяти аминокислот (пентафрагментов), начиная с N-конца белка, со специально созданной базой данных пентафрагментов белков, введенной в память компьютера и полученной с помощью специально написанных программ на основе файлов с координатами атомов структур белков из свободного доступа Protein Data Bank. На основе сведений о номерах папок, последовательно внесенных в рабочий файл для всех выделенных в последовательности аминокислот пентафрагментов, определяют положение α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре белка, по которым судят о вторичной структуре исследуемого белка. 18 табл.
Способ прогнозирования вторичной структуры белка на основе определения положения α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре белка, заключающийся в следующем:
A) создают базу данных аминокислотных пентафрагментов белков, содержащую папки с пентафрагментами, причем исходный список папок составлен по их названиям, сформированным на основании закодированного в двоичной системе описания водородных связей пептидных групп пентафрагментов во вторичной структуре белков, и записывают ее на информационный носитель;
Б) вводят в память компьютера записанную на информационный носитель базу данных аминокислотных пентафрагментов белков;
B) вводят в память компьютера программу FILEMAKER для представления информации о первичной структуре исследуемого белка в виде рабочего файла;
Г) вводят в память компьютера программу PREDICTOR для выделения пентафрагментов в рабочем файле исследуемого белка, поиска выделенных пентафрагментов в базе данных и записи названий папок базы данных, в которых обнаружены искомые пентафрагменты;
Д) вводят в память компьютера текстовый файл в виде
либо последовательности нуклеотидов, кодирующих исследуемый белок или его фрагмент;
либо последовательности аминокислот исследуемого белка или его фрагмента;
Е) текстовый файл представляют в виде рабочего файла, содержащего последовательность аминокислот исследуемого белка или его фрагмента с помощью ранее записанной в память компьютера программы FILEMAKER;
Ж) проводят поиск пентафрагментов исследуемого белка в базе данных с помощью ранее записанной в память компьютера программы PREDICTOR, при этом алгоритм программы включает в себя два этапа:
I) проведение поиска начального пентафрагмента, включающее
выделение в последовательности аминокислот исследуемого белка первого пентафрагмента;
запоминание и кодирование этого пентафрагмента с целью проведения поиска в базе данных;
проведение поиска первого пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы;
при нахождении первого пентафрагмента в базе данных на основе исходного списка папок считают этот фрагмент начальным и производят:
фиксирование номера папки базы данных, содержащей начальный пентафрагмент;
внесение номера папки базы данных, содержащей начальный пентафрагмент, в рабочий файл исследуемого белка;
при ненахождении первого пентафрагмента в базе данных на основе исходного списка папок производят:
сдвиг вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение следующего по порядку пентафрагмента;
запоминание и кодирование этого пентафрагмента с целью проведения поиска в базе данных;
проведение поиска следующего пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы;
повторение поиска начального пентафрагмента до нахождения искомого пентафрагмента в папках базы данных на основе исходного списка папок, введенного в текст программы, и назначение найденного пентафрагмента начальным;
II) проведение поиска последующих пентафрагментов после нахождения начального пентафрагмента, включающее
при совпадении начального пентафрагмента с первым пентафрагментом в последовательности аминокислот исследуемого белка производят:
сдвиг вперед вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска последующих пентафрагментов до конца последовательности аминокислот исследуемого белка;
- при несовпадении начального пентафрагмента с первым пентафрагментом в последовательности аминокислот исследуемого белка производят:
- сдвиг вперед вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
- запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
- создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
- проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
- фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска последующих пентафрагментов до конца последовательности аминокислот исследуемого белка;
- возврат к найденному начальному пентафрагменту;
- сдвиг назад вдоль последовательности аминокислот в рабочем файле исследуемого белка на одну аминокислоту и выделение нового пентафрагмента;
- запоминание и кодирование нового пентафрагмента с целью проведения поиска в базе данных;
- создание нового списка папок для поиска нового пентафрагмента на основе номера папки, содержащей ранее найденный пентафрагмент;
- проведение поиска нового пентафрагмента в базе данных на основе созданного списка папок;
фиксирование номера папки базы данных, содержащей найденный пентафрагмент;
- внесение номера папки базы данных, содержащей найденный пентафрагмент, в рабочий файл исследуемого белка;
- повторение поиска пентафрагмента до начала последовательности аминокислот исследуемого белка;
З) прогнозируют вторичную структуру белка по положению α-спиральных, β-структурных фрагментов и изгибов β-структуры в первичной структуре белка, определенному на основе сведений о номерах папок, последовательно внесенных в рабочий файл для всех пентафрагментов исследуемого белка или его фрагмента.
| WO 2004011909 A2, 05.02.2004 | |||
| БРАТУСЬ А.В | |||
| и др | |||
| Применение метода LOGIS для предсказания вторичной структуры белка | |||
| Биополимеры и клетка | |||
| Способ и аппарат для получения гидразобензола или его гомологов | 1922 |
|
SU1998A1 |
| БРАТУСЬ А.В | |||
| и др | |||
| Предсказания вторичной структуры белков модифицированным GUHA-методом | |||
| Биополимеры и клетка | |||
| Способ изготовления фанеры-переклейки | 1921 |
|
SU1993A1 |
Авторы
Даты
2011-08-10—Публикация
2010-04-09—Подача