Изобретение относится к компьютерному способу, использующему биохимические базы данных при разработке новых белковых соединений для фармацевтики, биотехнологии и других областей промышленности, а также для научных исследований в медицине, биохимии, молекулярной биологии и генетике, для которых существенно использование новых белковых соединений на основе аминокислот.
Данное изобретение относится к области белковой инженерии - направлению молекулярной биологии, в задачи которой входит создание знаний и методов, позволяющих получать белки с заранее заданной структурой и функцией. Одним из аспектов этого направления является проектирование (дизайн) белковых молекул. Задача проектирования является обратной по отношению к задаче прогнозирования белковой структуры. Если в процессе прогнозирования белковой структуры мы по известной нам последовательности аминокислот должны на первом этапе найти его вторичную структуру, т.е. положение α-спиральных, β-структурных участков и изгибов, то при проектировании мы должны задать такую ранее неизвестную последовательность аминокислот в первичной структуре, спроектированный нами для создания желаемой пространственной структуры, которая в подходящих условия, после ее синтеза примет порядок расположения и размер α-спиральных, β-структурных участков и изгибов.
Проектирование новых белков, как правило, осуществляется на основе разработанной методологии прогнозирования белковых структур и от успешности этой методологии зависит степень удачи при проектировании новых белков с заранее прогнозируемой структурой. В большинстве случаев полученные результаты - это лишь немногочисленные удачные примеры среди большого числа не упоминаемых авторами неудачных вариантов.
Известны попытки проектирования белковых структур, основанные на общих закономерностях их формирования. Одной из первых явилась работа группы Де Градо (D.Eisenberg, W.Wilcox, S.M.Eshita, P.M.Pryciak, S.P.Ho, W.F.Degrado. 1986. The design, synthesis, and crystallization of an alpha-helical peptide. Proteins: Structure, Function, and Bioinformatics. V.1, Issue 1, pp.16-22). Авторы исходили из простой идеи: гидрофобные взаимодействия белковых структур должны быть минимизированы и упрятаны в гидрофобное ядро, а гидрофильные - обеспечивать контакт с растворителем. Исходя из этих соображений авторы спроектировали и синтезировали искусственный белок, содержащий лишь несколько аминокислот (Leu, Glu, Lys) и состоящий из четырех α-спиралей (W.F.DeGrado, L.Regan, S.P.Но. The Design of a Four-helix Bundle Protein. Cold Spring Harb Symp Quant Biol 1987. 52: 521-526).
Однако такой упрощенный подход не позволяет проектировать близкие к реальным сложные белки, состоящие из 20 различных типов аминокислот и обладающие заданными как структурными, так и функциональными свойствами.
В основу искусственного белка альбебетина была положена не существующая в природе структура, которая состояла из двух повторов типа α-β-β (V.V.Chemeris, D.A.Dolgikh, A.N.Fedorov, A.V.Finkelstein, M.P.Kirpichnikov, V.N.Uversky, O.B.Ptitsyn. A new approach to artificial and modified proteins: theory-based design, synthesis in a cell-free system and fast testing of structural properties by radiolabels. Protein Eng. (1994) 7 (8): 1041-1052). Его структура была разработана на основе физической теории формирования вторичной структуры белков, развиваемой авторами (Ptitsyn O.B., Finkelstein A.V. Theory of protein secondary structure and algorithm of its prediction. Biopolymers. 1983. V.22. P.15-25). Структурное исследование альбебетина показало, что он обладает заданной авторами вторичной структурой и находится в состоянии расплавленной глобулы. Следует отметить, что точность подхода, используемого авторами, не превышает 80%, что не позволяет с полной уверенностью проектировать белки с заданной структурой. Авторы практически спроектировали лишь один белок, и далее исследования были прекращены.
С целью улучшения предсказательных свойств известного метода, использующего физические потенциалы, было предложено ввести ряд параметров, учитывающих свойства последовательностей аминокислот (A.M.Poole и R.Ranganathan. Knowledge-based potentials in protein design. Current Opinion in Structural Biology 2006, 16, 508-513). На основе этого метода, с учетом введенных параметров, авторы спроектировали de novo ряд белков (WO 2007030594, «Methods of using and analyzing biological sequence data», МПК G06F 19/22; G06F 19/18, опубл. 15.03.2007). Однако такой подход носить компилятивный характер и обеспечивает лишь некоторое улучшение взятых за основу методов, не меняя вероятностный характер исходного физического метода.
Известно изобретение, имеющее отношение к аппаратам и методам для количественного проектирования и оптимизации структуры белка (US 2002106694 «Apparatus and method for automated protein design», МПК С07К 1/00; C07K 14/00; C12N 15/10; G06F 17/50; G06F 19/00, опубл. 08.08.2002). Разработанный автоматический метод проектирования, количественно учитывающий взаимодействия поверхностных остатков боковых цепей на основе вычисления трех типов потенциалов и учета стереохимических ограничений, позволяет выбрать из большого числа вариантов белок FSD-1 с мотивом ββα, основанным на структуре домена цинк-фингер-белка. Последовательность аминокислот этого белка имеет очень малое сходство с этим доменом. Несмотря на это, исследование этого белка в растворе методом спектроскопии ядерного магнитного резонанса показали, что он образует структуру, полностью идентичную предложенному для нее дизайну (B.I.Dahiyat and S.L.Mayo. De Novo Protein Design: Fully Automated Sequence Selection. Science, (1997) 278, 82-87).
Недостатком этого метода является необходимость наличия образцового белка, на основе которого осуществляется выбор новой структуры из большого числа вариантов.
С помощью методологии Розетта (Rosetta), представленной в работе (Kuhlman В, Dantas G, Ireton GC, Varani G, Stoddard BL, Baker D. Design of a novel globular protein fold with atomic-level accuracy. Science, 2003, 302(5649), 1364-8), основанной на оптимизации выбранных структур, был спроектирован и синтезирован неизвестный в природе искусственный белок Тор 7, структура которого была подтверждена экспериментально. Ядро Rosetta - физическая модель макромолекулярных взаимодействий и алгоритмов поиска аминокислотной последовательности с наименьшей энергией для заданной белковой структуры. Авторы применили свой метод (US 7574306 «Method and system for optimization of polymer sequences with stable, 3-dimensional conformations», МПК G06F 19/00, опубл. 11.08.2009) к разработке конструкций ряда других белков. Однако этот метод требует довольно сложных расчетов и не всегда приводит к успешным результатам. Для его использования также необходимо наличие образцов.
Такие способы не решают проблемы создания простого способа проектирования новых белков, обладающих любой заданной структурой и функциональными свойствами, а необходимость использования в качестве образцов конкретных белковых структур ограничивает диапазон проектируемых структур.
Решение этой проблемы особенно важно в технологии изготовления фармацевтических и иммунологических препаратов белкового происхождения.
Задачей, на решение которой направлено заявляемое изобретение, является разработка способа проектирования первичной структуры белка, благодаря которому достигается технический результат, заключающийся в упрощении способа с расширением диапазона проектируемых структур.
Предлагаемый способ проектирования первичной структуры белка на основе получения характеризующей ее последовательности аминокислот и описания вторичной структуры, заключается в следующем:
A) создают базу данных аминокислотных пентафрагментов белков, содержащую папки с пентафрагментами, причем исходный список папок составлен по их названиям, сформированным на основании закодированного в двоичной системе описания водородных связей пептидных групп пентафрагментов во вторичной структуре белков, и записывают ее на информационный носитель;
Б) вводят в память компьютера записанную на информационный носитель базу данных аминокислотных пентафрагментов белков;
B) задают и вводят в память компьютера начальную последовательность из пяти аминокислот, принадлежащих к группе из двадцати канонических аминокислот белков, являющуюся заданным начальным пентафрагментом;
Г) задают и вводят в память компьютера описание вторичной структуры заданного начального пентафрагмента в виде десятизначного числа в двоичной системе;
Д) вводят в память компьютера программу PROTCOM для выделения и поиска пентафрагментов проектируемого белка в базе данных и записи названий аминокислот найденных пентафрагментов и номеров папок базы данных, описывающих вторичную структуру, в которых обнаружены искомые пентафрагменты;
Е) вводят и запоминают заданный начальный пентафрагмент проектируемого белка в виде последовательности из пяти аминокислот в программу PROTCOM;
Ж) вводят и запоминают заданную вторичную структуру заданного начального пентафрагмента в виде десятизначного числа в двоичной системе в программу PROTCOM;
З) проводят поиск заданного начального пентафрагмента проектируемого белка в базе данных с помощью ранее записанной в память компьютера программы PROTCOM, при этом алгоритм поиска включает в себя:
- кодирование заданного начального пентафрагмента для целей поиска в базе данных;
- проведение поиска заданного начального пентафрагмента в базе данных в папке с заданной вторичной структурой пентафрагмента;
- при нахождении в папке заданного начального пентафрагмента считают этот пентафрагмент первым из возможного числа N пентафрагментов проектируемой первичной структуры белка и производят:
- фиксирование номера папки базы данных, содержащей первый пентафрагмент;
- запись последовательности аминокислот первого пентафрагмента в рабочий файл программы;
- запись десятизначного номера папки, описывающего вторичную структуру найденного первого пентафрагмента в рабочий файл;
- при не нахождении в папке заданного начального пентафрагмента:
- задают и вводят в память компьютера новую начальную последовательность из пяти аминокислот, принадлежащих к группе из двадцати канонических аминокислот белков, являющуюся новым заданным начальным пентафрагментом;
- вводят и запоминают новый заданный начальный пентафрагмент проектируемого белка в виде последовательности из пяти аминокислот в программу PROTCOM;
- проводят поиск нового заданного начального пентафрагмента проектируемого белка в базе данных с помощью ранее записанной в память компьютера программы PROTCOM, при этом алгоритм поиска включает в себя:
- кодирование нового заданного начального пентафрагмента для целей поиска в базе данных;
- проведение поиска нового заданного начального пентафрагмента в базе данных в папке с заданной вторичной структурой пентафрагмента;
- повторение задания новых начальных пентафрагментов и поиска новых заданных исходных пентафрагментов осуществляют до тех пор, пока не будет найден пентафрагмент с такой последовательностью аминокислот, которая находится в папке базы данных, описывающей заданную вторичную структуру пентафрагмента;
И) задают вторичные структуры каждого последующего из (N-1) пентафрагментов путем введения того же или измененного десятизначного числа, описывающего вторичную структуру предыдущего пентафрагмента в программу PROTCOM;
К) проводят поиск в базе данных пентафрагментов, содержащих четыре аминокислоты каждого из (N-1) пентафрагментов, записанных в рабочем файле и одну новую, при этом алгоритм поиска включает в себя:
- выделение и запоминание четырех последних аминокислот в каждом из (N-1) пентафрагментов, записанных в рабочем файле;
- поиск пентафрагментов, содержащих четыре последние аминокислоты каждого из (N-1) пентафрагментов, записанных в рабочем файле, и одну новую аминокислоту в базе данных в папке с заданной вторичной структурой;
- при нахождении таких пентафрагментов производят:
- выбор одной из новых аминокислот и присоединение ее к четырем последним аминокислотам предыдущего пентафрагмента;
- запись новой аминокислоты в рабочий файл, отражающий проектируемую первичную структуру белка;
- запись десятичного номера папки, описывающего вторичную структуру каждого найденного пентафрагмента;
- при не нахождении таких пентафрагментов производят:
- задание измененной вторичной структуры;
- выделение четырех последних аминокислот в последующем пентафрагменте;
- поиск пентафрагментов, содержащих четыре последних аминокислоты предыдущего пентафрагмента и одну новую аминокислоту в базе данных в папке с измененной вторичной структурой;
- повторение изменения вторичной структуры и поиск в базе данных осуществляют до тех пор, пока не будет найден хотя бы один пентафрагмент, содержащий четыре аминокислоты предыдущего пентафрагмента;
Л) спроектированной первичной структурой белка считают полученную в рабочем файле последовательность аминокислот, с соответствующим описанием ее вторичной структуры.
Способ осуществляют следующим образом:
А) создают базу данных аминокислотных пентафрагментов белков, содержащую папки с пентафрагментами, причем исходный список папок составлен по их названиям, сформированным на основании закодированного в двоичной системе описания водородных связей (Н-связей) пептидных групп пентафрагментов во вторичной структуре белков, и записывают ее на информационный носитель;
а) из Protein Data Bank производят скачивание находящихся в открытом доступе файлов с координатами атомов кристаллов белков, исследованных методом рентгеноструктурного анализа (РСА). Для создания начальной базы было произведено скачивание 2500 файлов белков.
б) с помощью компьютерной программы Protein 3D (Компьютерная программа «Protein 3D», зарегистрировано в Рос. АПО, №980143 от 03.05.98, авторы: Карасев В.А., Демченко Е.Л.) на основе полученных из Protein Data Bank файлов создают текстовые файлы, содержащие первичные структуры белков с описанием Н-связей, образуемых пептидными группами основных цепей белков во вторичной структуре;
в) с помощью комплекса программ для создания базы проводят следующие действия:
- осуществляют нарезку полученных первичных структур белков на фрагменты из пяти аминокислот (пентафрагменты) таким образом, чтобы каждый последующий фрагмент в процессе движения снизу вверх выделялся со сдвигом на одну аминокислоту по отношению к предыдущему фрагменту, а информация об Н-связях каждого выделяемого фрагмента во вторичной структуре белка полностью сохранялась. В таблице 1 для примера показана процедура нарезки фрагмента текстового файла белка 1SCN (субтилизина Карлсберга). Из таблицы видно, что Н-связи в пентафрагментах остаются неизменными.
- пентафрагменты, гомологичные по структуре Н-связей пептидных групп во вторичной структуре белка, сортируют по папкам, присваивая названиям папок закодированное в двоичной системе описание Н-связей пептидных групп. Наличие Н-связи обозначают цифрой «1», отсутствие водородной связи - цифрой «0».
В каждом пентафрагменте имеется 5 пар пептидных групп, Н-связи связи которых описываются четырьмя видами пар переменных: нет Н-связей - 00, Н-связь O…HN - 01, Н-связь NH…О - 10 и две Н-связи: О…HN и NH…O - 11. Таким образом название папки, содержащей гомологичные по структуре пентафрагменты, состоит из 10 символов 0 и 1, прочитываемых сверху вниз и записываемых в строку слева направо.
Примеры вариантов выделяемых пентафрагментов и описывающих их десятизначных чисел в двоичной системе приведены в таблице 2. Так, пентафрагмент, полученный из участка β-структуры (первая строка, пример слева), не содержит Н-связей ближнего порядка и описывается числом 0000000000. Участок с пентафрагментом, который находится в переходной области β-структура - α-спираль (первая строка, пример справа) содержит одно звено со связями O…HN и NH…O - пара переменных 11 и четыре звена со связями O…HN - 01 и характеризуется числом 1101010101. Центральная область α-спирали, как показано в таблице 2, содержит пять звеньев со связями O…HN и NH…O - 11 и описывается числом 1111111111. Переходная область α-спираль - β-структура содержит четыре звена со связями NH…O - 10 и одно - со связями O…HN и NH…O - пара переменных 11, что дает десятизначное число 1010101011. Наконец, участок изгиба β-структуры с одной Н-связью, как следует из таблицы 2, содержит одно звено со связью NH…O - 10, три звена - без Н-связей - 00 и одно звено со связью O…HN - 01, что описывается числом 1000000001.
При создании базы данных в процессе обработки текстовых файлов производилось движение по цепи белка снизу вверх со сдвигом на одну аминокислоту на каждом этапе и при этом каждый выделяемый пентафрагмент получал соответствующее десятизначное описание. В таблице 1 эти значения приведены во втором справа столбце. В результате в этом столбце мы имеем серии перекрывающихся на 4/5 десятизначных описаний структуры участка белка 1CSN, каждое из которых получает в базе данных папку с аналогичным номером. Жирным шрифтом выделены 10-значные номера, для пентафрагментов, аналогичных приведенным в таблице 2.
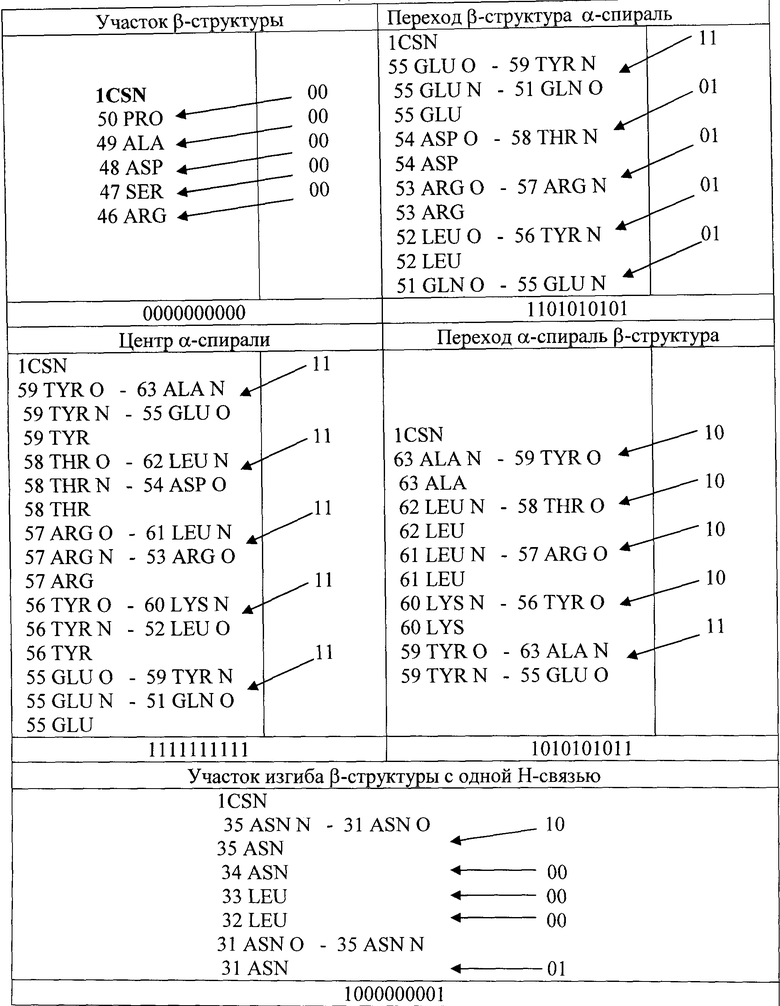
Центральные участки α-спиралей и β-структур белков описывают, соответственно, ряды повторяющихся 10-значных номеров 1111111111 и 0000000000. В то же время переходные участки от β-структуры к α-спирали и от α-спирали к β-структуре описываются блоками 10-значных номеров с постепенно изменяющимся составом пар переменных. Примеры таких блоков приведены в таблице 3. Жирным шрифтом выделены начальные и конечные участки переходов и их 10-значные описания.
58 THR O - 62 LEU N
Нами было установлено, что количество таких блоков ограничено и между переходами от β-структуры к α-спирали и от α-спирали к β-структуре имеется антисимметрия (0←→1). Для этих переходов составлен каталог. Аналогичные антисимметрии (0←→1) наблюдаются также для изгибов α-спиралей и β-структур, примеры которых представлены в таблице 4. Для этих блоков также составлен каталог. Жирным шрифтом выделены начало и конец изгибов и пары переменных, обозначающие водородные связи в изгибах.
Путем комбинации эти блоки могут быть использованы для проектирования любых типов вторичных структур белков.
г) производят упрощение выделенных пентафрагментов путем удаления из них информации о структуре Н-связей и оставления только последовательности из пяти аминокислот;
д) с целью облегчения дальнейшей процедуры поиска пентафрагментов в базе данных производят их сортировку на файлы, содержащие фрагменты с одинаковым пятизначным числовым индексом, который им присваивают путем отнесения каждой из аминокислот пентафрагмента к одной из четырех групп преобразований антисимметрии (Карасев В.А., Лучинин В.В. Введение в конструирование бионических наносистем. - М.: Физматлит, 2009, 464 с., глава 8). Эти группы приведены в таблице 5.
При этом в имени файла записывают пятизначный индекс и название папки, в которой этот файл расположен. Если пентафрагмент
описывается 10-значным числом 0000000000, его индекс формируют сверху вниз и записывают слева направо: например, если аминокислота Efg относится к группе 1, Def -к группе 2, Cde - к группе 3, Bcd - к группе 4 и Abc - к группе 1, то его 5-значный индекс будет 12341, а имя файла - 12341_0000000000.
Созданная база данных содержит более 500 тысяч пентафрагментов, сортированных на более чем 500 папок. База данных организована в систему, состоящую из 16 гиперкубов, изоморфных булевым гиперкубам В6 (База данных пентафрагментов белков. Авторы: В.А.Карасев, А.И.Беляев, В.В.Лучинин. Зарегистрирована 7 июля 2010 года в Федеральном агентстве РОСПАТЕНТ №2010620364).
База данных может постоянно пополняться путем обработки новых файлов из Protein Data Bank. Также может быть создана теоретическая база данных.
Б) вводят в память компьютера записанную на информационный носитель базу данных аминокислотных пентафрагментов белков;
В) задают и вводят в память компьютера начальную последовательность из пяти аминокислот, принадлежащих к группе из двадцати канонических аминокислот белков, являющуюся заданным начальным пентафрагментом;
Задуманная начальная последовательность из пяти аминокислот представлена в виде столбца из трехбуквенных сокращенных названий аминокислот с обозначениями слева их номеров, записанная снизу вверх:
Г) задают и вводят в память компьютера описание вторичной структуры заданного начального пентафрагмента в виде десятизначного числа в двоичной системе;
Д) вводят в память компьютера программу PROTCOM для выделения и поиска пентафрагментов проектируемого белка в базе данных и записи названий аминокислот найденных пентафрагментов и номеров папок базы данных, описывающих вторичную структуру, в которых обнаружены искомые пентафрагменты;
Е) вводят и запоминают заданный начальный пентафрагмент проектируемого белка в виде последовательности из пяти аминокислот в программу PROTCOM;
Оператор вводит в программу задуманную последовательность из пяти аминокислот (заданный начальный пентафрагмент).
Ввод этих аминокислот в программу осуществляется сверху вниз, начиная с пятой аминокислоты, и заканчивается первой аминокислотой: Efg, Def, Cde, Bcd, Abc.
Ж) вводят и запоминают заданную вторичную структуру заданного начального пентафрагмента в виде десятизначного числа в двоичной системе в программу PROTCOM;
Пример вводимого десятизначного числа: 0000000000
З) проводят поиск заданного начального пентафрагмента проектируемого белка в базе данных с помощью ранее записанной в память компьютера программы PROTCOM, при этом алгоритм поиска включает в себя:
- кодирование заданного начального пентафрагмента для целей поиска в базе данных;
Программа считывает аминокислоты пентафрагмента сверху вниз, кодирует их в соответствии с принадлежностью к той или иной группе антисимметрии и записывает кодовый номер слева направо, аналогично сформированным индексам файлов, например: Efg - 1, Def - 2, Cde - 3, Bсd - 4, Abс - 4, кодовый номер - 12344.
- проведение поиска заданного начального пентафрагмента в базе данных в папке с заданной вторичной структурой пентафрагмента;
Для введенного десятизначного числа 0000000000 заданный начальный пентафрагмент ищут в папке базы данных с номером 0000000000, в файле с кодовым номером 12344, т.е. 12344_0000000000.
- при нахождении в папке заданного начального пентафрагмента считают этот пентафрагмент первым из возможного числа N пентафрагментов проектируемой первичной структуры белка и производят:
- фиксирование номера папки базы данных, содержащей первый пентафрагмент;
- запись последовательности аминокислот первого пентафрагмента в рабочий файл программы;
- запись десятизначного номера папки, описывающего вторичную структуру найденного первого пентафрагмента в рабочий файл;
Формат рабочего файла, создаваемого программой PROTCOM, показан в таблице 6.
Запись последовательности аминокислот исследуемого белка в рабочем файле производится снизу вверх, что отражает порядок синтеза белка на рибосоме (удлинение белка происходит путем добавления аминокислот к верхней аминокислоте). Столбцы файла имеют следующее назначение:
1 - номера аминокислот в проектируемом белке, записанные снизу вверх;
2 - последовательность аминокислот в проектируемом белке, записанная снизу вверх с помощью трехбуквенных обозначений;
3 - десятизначные номера папок (bbbbbbbbbb) базы данных, описывающих вторичную структуру проектируемых пентафрагментов, записанные снизу вверх.
в строке N - сигнальное значение конца белковой последовательности (STP).
Жирным шрифтом выделен первый пентафрагмент и десятизначный номер папки, в которой найден данных пентафрагмент.
- при не нахождении в папке заданного начального пентафрагмента:
- задают и вводят в память компьютера новую начальную последовательность из пяти аминокислот, принадлежащих к группе из двадцати канонических аминокислот белков, являющуюся новым заданным начальным пентафрагментом;
- вводят и запоминают новый заданный начальный пентафрагмент проектируемого белка в виде последовательности из пяти аминокислот в программу PROTCOM;
- проводят поиск нового заданного начального пентафрагмента проектируемого белка в базе данных с помощью ранее записанной в память компьютера программы PROTCOM, при этом алгоритм поиска включает в себя:
- осуществляют кодирование нового заданного начального пентафрагмента для целей поиска в базе данных;
- проводят поиск нового заданного начального пентафрагмента в базе данных в папке с заданной вторичной структурой пентафрагмента;
- повторение задания новых начальных пентафрагментов и поиска новых заданных начальных пентафрагментов осуществляют до тех пор, пока не будет найден пентафрагмент с такой последовательностью аминокислот, которая находится в папке базы данных, описывающей заданную вторичную структуру пентафрагмента.
И) задают вторичные структуры каждого последующего из (N-1) пентафрагментов, записанных в рабочем файле путем введения того же или измененного десятизначного числа, описывающего вторичную структуру предыдущего пентафрагмента, в программу PROTCOM;
К) проводят поиск в базе данных пентафрагментов, содержащих четыре аминокислоты каждого из (N-1), записанных в рабочем файле пентафрагментов, и одну новую, при этом алгоритм поиска включает в себя:
- выделение и запоминание четырех последних аминокислот в каждом из (N-1) пентафрагментов, записанных в рабочем файле;
- поиск пентафрагментов, содержащих четыре последние аминокислоты каждого из (N-1) пентафрагментов, записанных в рабочем файле, и одну новую аминокислоту в базе данных в папке с заданной вторичной структурой;
Например, в таблице 7 жирным шрифтом выделены четыре последние аминокислоты предыдущего пентафрагмента и введенное описание вторичной структуры для поиска нового пентафрагмента.
- при нахождении таких пентафрагментов производят:
- выбор одной из новых аминокислот и присоединение ее к четырем последним аминокислотам предыдущего пентафрагмента;
- производят запись новой аминокислоты в рабочий файл, отражающий проектируемую первичную структуру белка;
- производят запись десятичного номера папки, описывающего вторичную структуру каждого найденного пентафрагмента;
- при не нахождении таких пентафрагментов производят:
- задание измененной вторичной структуры;
- выделение четырех последних аминокислот в последующем пентафрагменте;
- поиск пентафрагментов, содержащих четыре последних аминокислоты предыдущего пентафрагмента и одну новую аминокислоту в базе данных в папке с измененной вторичной структурой;
- повторение изменения вторичной структуры и поиск в базе данных осуществляют до тех пор, пока не будет найден хотя бы один пентафрагмент, содержащий четыре аминокислоты предыдущего пентафрагмента;
Л) считают полученную в рабочем файле последовательность аминокислот с соответствующим описанием ее вторичной структуры спроектированной первичной структурой белка.
В результате действий программы PROMCOM и работы оператора, проектирующего белок, в рабочем файле оказывается полностью заполненным второй столбец, содержащий первичную структуру белка и третий столбец, на основе которого судят о вторичной структуре этого белка. Наличие в 3-м столбце идущих подряд папок 0000000000 характеризует фрагмент как β-структурный. Несколько идущих подряд папок с нумерацией 1111111111 позволяет отнести фрагмент к α-спиральному (см. таблицу 2). Переходные участки между α-спиральной и β-структурной конформации, а также изгибы β-структуры (таблицы 2-4) проектируются и описываются соответствующими папками.
Описание заявки иллюстрируют следующие графические материалы:
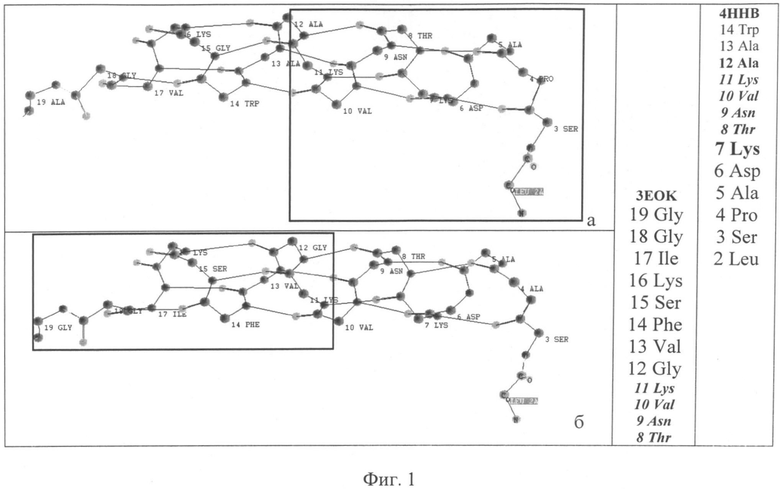
Фиг.1. Скринпринты фрагментов вторичной структуры белков 4ННВ и 3EOK, полученные с помощью программы PROTEIN 3D.
а - 4ННВ (человек); б - 3ЕОК (утка);
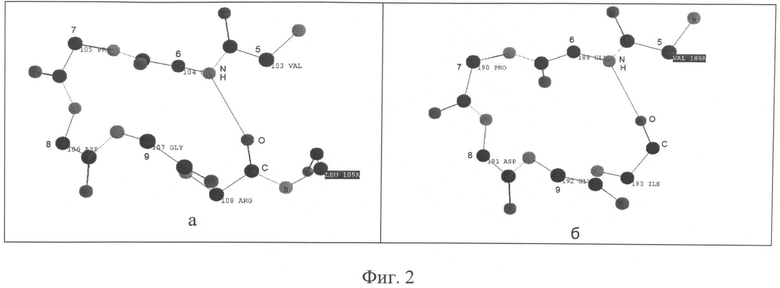
Фиг.2. Скринпринты фрагментов вторичной структуры белков 1AGD и 2R37, полученных с помощью программы PROTEIN 3D для проектируемого участка белка.
а - пентафрагмент 1AGD (103-107); б - пентафрагмент 2R37 (189-193).
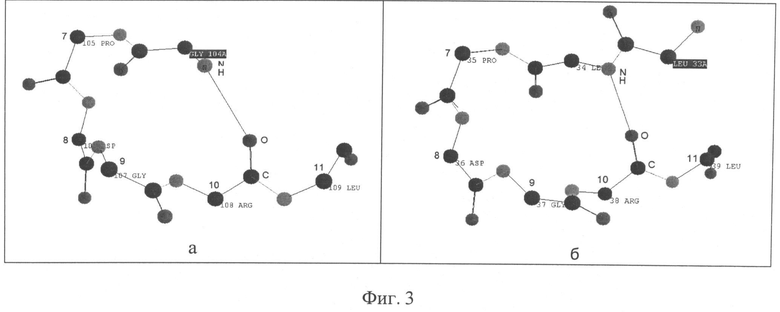
Фиг.3. Скринпринты фрагментов вторичной структуры белков 1AGD и 3В02, полученных с помощью программы PROTEIN 3D для проектируемого участка белка.
а - пентафрагмент 1AGD (105-109); б - пентафрагмент 3В02 (35-39).
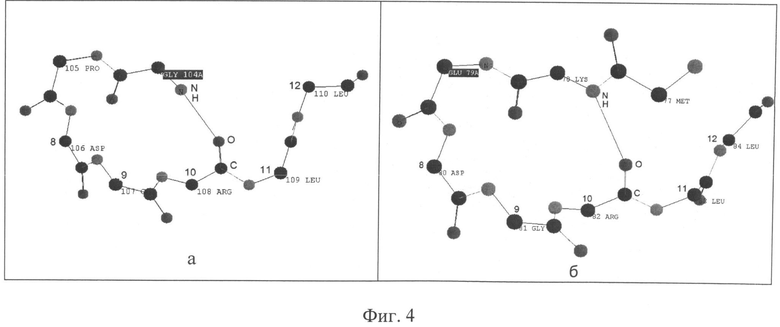
Фиг.4. Скринпринты фрагментов вторичной структуры белков 1AGD и 1BAS, полученных с помощью программы PROTEIN 3D для проектируемого участка белка.
а - пентафрагмент 1AGD (106-110); б - пентафрагмент 1BAS (80-84).
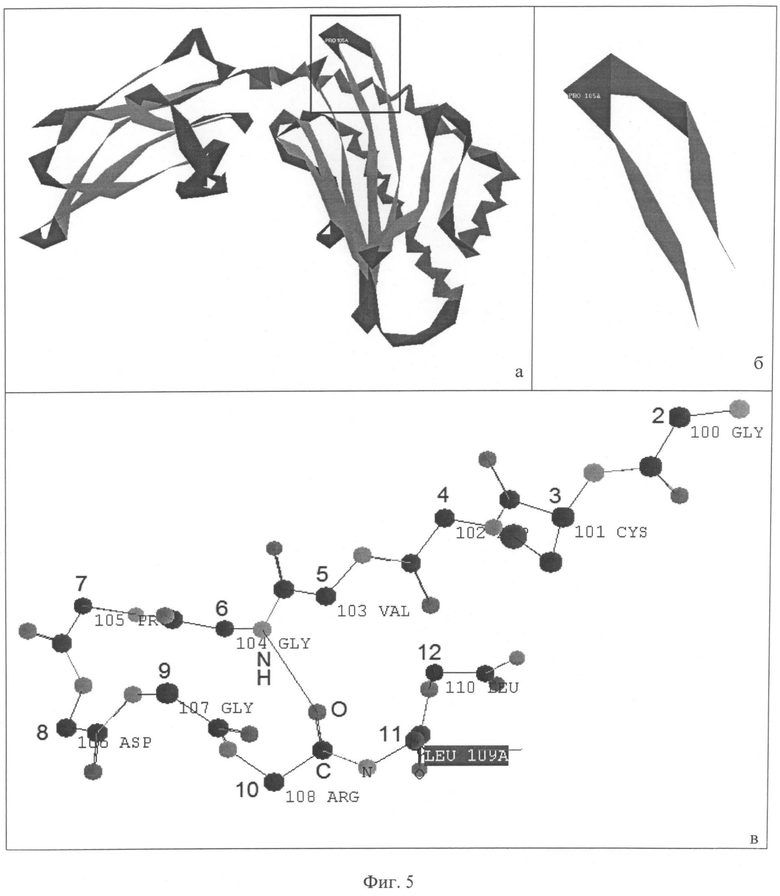
Фиг.5. Скринпринты белка 1AGD, полученные с помощью программы PROTEIN 3D для белка 1AGD, исследованного методом PCA.
а - общий вид белка; б - вид фрагмента белка; в - детальный вид вторичной структуры белка 1GDJ, соответствующий заданной вторичной структуре примера 2.
Способ поясняется примерами.
Пример 1.
В данном примере рассмотрен способ проектирования первичной структуры белка, с заданной в виде α-спирали вторичной структурой, содержащей участок перехода от β-структуры к α-спирали, центральную область α-спирали и участок перехода от α-спирали к β-структуре.
При проведении способа проектирования первичной структуры белка с заданной вторичной структурой на основе получения характеризующей ее последовательности аминокислот и описания вторичной структуры, осуществляют следующее:
A) создают базу данных аминокислотных пентафрагментов белков, содержащую папки с пентафрагментами, причем исходный список папок составлен по их названиям, сформированным на основании закодированного в двоичной системе описания водородных связей пептидных групп пентафрагментов во вторичной структуре белков, и записывают ее на информационный носитель;
Б) создают каталог описаний вторичных структур, содержащий описания вторичных структур в виде последовательности десятизначных булевых чисел;
B) вводят в память компьютера записанную на информационный носитель базу данных аминокислотных пентафрагментов белков;
Г) задают описание вторичной структуры проектируемой первичной структуры белка в виде последовательности десятизначных булевых чисел на основе каталога описаний вторичных структур;
В данном примере заданная в виде α-спирали вторичная структура содержит участки перехода от β-структуры к α-спирали, центральную область α-спирали и участки перехода от α-спирали к β-структуре. Их описание оператор находит в каталоге вторичных структур и фиксирует его (табл.8).
Д) задают и вводят в память компьютера начальную последовательность из пяти аминокислот, принадлежащих к группе из двадцати канонических аминокислот белков, являющуюся заданным начальным пентафрагментом:
которая записана в порядке номеров снизу вверх Leu, Ser, Pro, Ala, Asp.
E) задают и вводят в память компьютера описание вторичной структуры заданного начального пентафрагмента в виде десятизначного числа в двоичной системе, являющегося первым десятизначным числом в заданном описании вторичной структуры, которое соответствует названию папки в базе данных, содержащей указанный начальный пентафрагмент: десятизначное число 0101010100. Как видно из таблицы 8, оно является первым десятизначным числом в заданном описании вторичной структуры.
Оно соответствует описанию пентафрагмента, имеющего четыре Н-связи C=O…HN, описываемых парой переменных 01, и одну пару переменных 00 (нет Н-связей) - см. таблицу 3 (переход β-структура - α-спираль).
Ж) вводят в память компьютера программу PROTCOM для выделения и поиска пентафрагментов проектируемого белка в базе данных и записи названий аминокислот найденных пентафрагментов и номеров папок базы данных, описывающих вторичную структуру, в которых обнаружены искомые пентафрагменты;
1. Установка программы проводится в специальную папку, в которую продуцируются рабочие файлы, содержащие проектируемую первичную структуру белка и описывающую ее вторичную структуру в двоичной системе десятизначных чисел.
2. Вновь установленная программа не содержит никаких других файлов кроме самой программы и открывается заставкой для ввода заданного начального пентафрагмента.
3. В начале программы приведена в виде таблицы система двадцати аминокислот, состоящая из четырех групп.
З) вводят и запоминают заданный начальный пентафрагмент проектируемого белка в виде последовательности из пяти аминокислот в программу PROTCOM: оператор осуществляет ввод аминокислот, составляющих заданный начальный пентафрагмент в последовательности с пятой по первую, т.е. сверху вниз: Asp, Ala, Pro, Ser, Leu.
И) вводят и запоминают заданное описание вторичной структуры заданного начального пентафрагмента в виде десятизначного числа в двоичной системе в программу PROTCOM: оператор вводит в программу PROTCOM последовательность 0101010100.
К) проводят поиск заданного начального пентафрагмента проектируемого белка в базе данных с помощью ранее записанной в память компьютера программы PROTCOM, при этом алгоритм поиска включает в себя:
- кодирование заданного начального пентафрагмента для целей поиска в базе данных;
Кодирование осуществляется программой путем отнесения каждой из аминокислот заданного начального пентафрагмента к той или иной группе антисимметрии (табл.5 описания заявки).
В данном примере: Asp - 4, Ala - 2, Pro - 1, Ser - 3, Leu - 2. Эта числовая последовательность записывается в память программы слева направо 42132 и используется для поиска заданного начального пентафрагмента в папке 0101010100 базы данных в файле 42132_0101010100.
- проведение поиска заданного начального пентафрагмента в базе данных в папке с заданным описанием вторичной структуры пентафрагмента;
Программа обнаружила заданный начальный пентафрагмент в файле 42132_0101010100:
Этот пентафрагмент был выделен из текстового файла, полученного программой PROTEIN 3D на основе обработки координат атомов белка из Protein Data Bank, и имеет структуру 0101010100 переходного участка β-структура - α-спираль (см. таблицу 8).
Таблицы 9, 10, 11 иллюстрируют работу программы. В левой части, озаглавленной «Ввод», размещаются: в первом столбце вводимые в программу PROTCOM порядковые номера проектируемых аминокислот, во втором столбце помещаются аминокислоты при вводе согласно п.З) или пары переменных согласно п.Л), выбираемые оператором на основе заданной вторичной структуры (таблицы 8). В третьем столбце записывается введенное в программу описание вторичной структуры в виде десятизначного числа. В центральной части, озаглавленной «Поиск пентафрагмента в базе данных», помещаются: в первом столбце наименования файлов с номером кодировки и номером заданной папки, а во втором - наименования найденных в пентафрагментах аминокислот. В правой части таблиц проводится запись, осуществляемая программой PROTCOM в рабочем файле после обнаружения заданного начального пентафрагмента, а в дальнейшем - после выбора аминокислоты в файле с номером кодировки номером заданной папки.
- при нахождении в папке заданного начального пентафрагмента считают этот пентафрагмент первым из возможного числа N пентафрагментов проектируемой первичной структуры белка и производят:
- фиксирование номера папки базы данных, содержащей первый пентафрагмент;
- запись последовательности аминокислот первого пентафрагмента в рабочий файл программы;
- запись десятизначного номера папки, описывающего вторичную структуру найденного первого пентафрагмента в рабочий файл;
Программа обнаружила введенный начальный пентафрагмент в файле с соответствующей кодировкой и номером папки, и делает запись в рабочем файле (табл.9).
Поскольку заданный начальный пентафрагмент был найден, то мы опускаем действия поиска, относящиеся к случаю не нахождения в папке заданного начального пентафрагмента.
Л) задают описание вторичной структуры для каждого последующего из (N-1) пентафрагментов, используя описание заданной вторичной структуры в виде последовательности десятизначных булевых чисел, которые соответствуют названиям папок в базе данных, содержащих указанные пентафрагменты, путем введения того же или измененного десятизначного числа, описывающего вторичную структуру предыдущего пентафрагмента, в программу PROTCOM;
Для этого в процессе задания описания вторичной структуры программа PROTCOM предлагает ввести пары переменных 00, 01, 10 или 11. Из таблицы 8 видно, что следующим десятизначным числом является 1101010101. По этой причине оператор выбирает 11, и вводит пару переменных 11 в программу (столбец «Аминокислоты и пары переменных» в таблице 10). Программа добавляет 11 слева и удаляет пару цифр справа, что приводит к изменению десятизначного числа, описывающего вторичную структуру предыдущего пентафрагмента, что отражено в столбце «Описание заданной вторичной структуры» таблицы 10.
M) проводят поиск в базе данных пентафрагментов, содержащих четыре аминокислоты каждого из (N-1) пентафрагментов, записанных в рабочем файле, и одну новую, при этом алгоритм поиска включает в себя:
- выделение и запоминание четырех последних аминокислот в каждом из (N-1) пентафрагментов, записанных в рабочем файле;
- поиск пентафрагментов, содержащих четыре последние аминокислоты каждого из (N-1) пентафрагментов, записанных в рабочем файле, и одну новую аминокислоту в базе данных в папке с заданным описанием вторичной структуры;
Для этого программа выделяет в пентафрагменте, записанном рабочем файле таблицы 10, четыре аминокислоты, записанных сверху вниз: Asp, Ala, Pro, Ser.
Далее программа их кодирует в соответствии с принадлежностью к той или иной группе антисимметрии и записывает кодовый номер слева направо, аналогично сформированным индексам файлов, но без первой аминокислоты: 4213 и проводит в базе данных поиск пентафрагментов, содержащих четыре выделенных аминокислоты, в папке с заданной структурой следующего пентафрагмента (1101010101), т.е в файлах Х4213_1101010101, где X может принимать значения 1, 2, 3, 4, соответствующие номерам групп антисимметрии (см. таблицу 5 описания заявки) - 14213_1101010101, 24213_1101010101, 34213_1101010101, 44213_1101010101.
В результате поиска были найдены пентафрагменты, содержащие четыре последних аминокислоты: Asp, Ala, Pro, Ser и следующие пятые аминокислоты, записанные вместе с кодами белков, из которых они были получены:
- в файле группы 1 (14213_1101010101): пентафрагменты не найдены;
- в файле группы 2 (24213_1101010101) - пентафрагменты не найдены;
- в файле группы 3 (34213_1101010101): 1 - Ser;
- в файле группы 4 (44213_1101010101): 2 - Lys;
- при нахождении таких пентафрагментов оператор производит:
- выбор одной из новых аминокислот и присоединение ее к четырем последним аминокислотам предыдущего пентафрагмента;
Из найденных программой аминокислот можно выбрать либо Ser в файле группы 3, либо Lys в файле группы 4. При этом программа допускает выбор только одного варианта. В зависимости от выбора результат проектирования будет различным, что может быть выяснено только в результате проектирования. В качестве пятой аминокислоты оператор выбрал Lys и ввел эту информацию в программу.
Далее программа производит:
- запись новой аминокислоты в рабочий файл («Запись в рабочем файле», табл.10), отражающий проектируемую первичную структуру белка (Lys);
- запись десятичного номера папки, описывающего вторичную структуру каждого найденного пентафрагмента (1101010101);
Поскольку пентафрагмент был найден, то мы опускаем этапы поиска, относящиеся к случаю не нахождения в папке пентафрагмента.
Далее производят повторение действий согласно пп. Л) и М) до окончания процесса проектирования. Как видно из таблицы 11, в процессе проектирования последовательности аминокислот на этапах 11, 13 и 18 появлялась возможность выбора из двух или трех аминокислот, на остальных этапах программа обнаруживала лишь одну аминокислоту.
Н) спроектированной первичной структурой белка считают полученную в рабочем файле последовательность аминокислот, с соответствующим описанием ее вторичной структуры, записанная в рабочем файле программы PROTCOM и представленная в правой части таблицы 11.
Для экспериментального подтверждения в существовании спроектированной в примере 1 первичной структуры белка с заданной вторичной структурой в Protein Data Bank было найдено несколько фрагментов белков, имеющих последовательность аминокислот, частично совпадающую с последовательностью аминокислот, представленную в примере 1 (табл.12).
Как видно из таблицы 12, спроектированная первичная структура белка в примере 1 имеет наибольшее сходство с первичной структурой фрагментов белков 4ННВ и 3ЕОК. Так, спроектированная первичная структура белка с 1-го по 10-ю аминокислоту полностью идентична с первичной структурой фрагмента белка 4ННВ со 2-й по 11-ю аминокислоту. В то же время, с 11-й по 18-ю аминокислоты спроектированная первичная структура белка идентична с первичной структурой фрагмента белка 3ЕОК с 12-й по 19-ю аминокислоты.
В таблице 13 приведено двумерное описание водородных связей представленных в таблице 12 фрагментов белков, полученное с помощью программы Protein 3D на основе файлов таблицы 12. Там же дано описание их вторичной структуры в виде десятизначных булевых чисел, которое полностью идентично заданному описанию вторичной структуры спроектированной первичной структуры (таблица 11).
На фиг.1,а и 1,б представлены скринпринты фрагментов вторичной структуры белков 4ННВ и 3EOK, полученные с помощью программы Protein 3D, и соответствующие им последовательности аминокислот первичной структуры. На этих фигурах видно, что вторичная структура фрагментов, из которых состоит спроектированная первичная структура примера 1, имеет перекрывание с 7-й по 11-ю аминокислоты, так же как и перекрывание последовательностей аминокислот их первичной структуры (на последовательностях оно выделено курсивом). Следовательно, спроектированная последовательность имеет идентичную с исходными фрагментами вторичную структуру.
Таким образом, в примере 1 представлена спроектированная первичная структура белка, состоящая из фрагментов белков 4ННВ и 3ЕОК, заданная вторичная структура которой полностью совпадает с вторичной структурой каждого из этих белков.
Сведения о вторичной структуре белков 4ННВ и 3EOK, относящихся к классу гемоглобинов, опубликованы и представлены таблице 14.
Пример 2.
В данном примере рассмотрен способ проектирования первичной структуры белка, с заданной в виде инвертированного β-изгиба вторичной структурой.
При проведении способа проектирования первичной структуры белка с заданной вторичной структурой на основе получения характеризующей ее последовательности аминокислот и описания вторичной структуры, осуществляют следующее:
A) создают базу данных аминокислотных пентафрагментов белков, содержащую папки с пентафрагментами, причем исходный список папок составлен по их названиям, сформированным на основании закодированного в двоичной системе описания водородных связей пептидных групп пентафрагментов во вторичной структуре белков, и записывают ее на информационный носитель;
Б) создают каталог описаний вторичных структур, содержащий описания вторичных структур в виде последовательности десятизначных булевых чисел;
B) вводят в память компьютера записанную на информационный носитель базу данных аминокислотных пентафрагментов белков;
Г) задают описание вторичной структуры проектируемой первичной структуры белка в виде последовательности десятизначных булевых чисел на основе каталога описаний вторичных структур;
В данном примере вторичная структура задана в виде инвертированного β-изгиба. Ее описание оператор находит в каталоге вторичных структур и фиксирует ее (табл.15).
Д) задают и вводят в память компьютера начальную последовательность из пяти аминокислот, принадлежащих к группе из двадцати канонических аминокислот белков, являющуюся заданным начальным пентафрагментом:
которая записана в порядке номеров снизу вверх: Tyr, Gly, Cys, Asp, Val.
E) задают и вводят в память компьютера описание вторичной структуры заданного начального пентафрагмента в виде десятизначного числа в двоичной системе, являющегося первым десятизначным числом в заданном описании вторичной структуры, которое соответствует названию папки в базе данных, содержащей указанный начальный пентафрагмент: 10-значное число 0000000000. Как видно из таблицы 15, оно является первым десятизначным числом в заданном описании вторичной структуры.
Ж) вводят в память компьютера программу PROTCOM для выделения и поиска пентафрагментов проектируемого белка в базе данных и записи названий аминокислот найденных пентафрагментов и номеров папок базы данных, описывающих вторичную структуру, в которых обнаружены искомые пентафрагменты;
Установка программы проводится аналогично п.Ж в примере 1.
З) вводят и запоминают заданный начальный пентафрагмент проектируемого белка в виде последовательности из пяти аминокислот в программу PROTCOM: оператор осуществляет ввод аминокислот, составляющих заданный начальный пентафрагмент в последовательности с пятой по первую, т.е. сверху вниз: Val, Asp, Cys, Gly, Tyr.
И) вводят и запоминают заданное описание вторичной структуры заданного начального пентафрагмента в виде десятизначного числа в двоичной системе в программу PROTCOM: оператор вводит в программу PROTCOM последовательность 0000000000.
К) проводят поиск заданного начального пентафрагмента проектируемого белка в базе данных с помощью ранее записанной в память компьютера программы PROTCOM, при этом алгоритм поиска включает в себя:
- кодирование заданного начального пентафрагмента для целей поиска в базе данных;
Кодирование осуществляется программой путем отнесения каждой из аминокислот заданного начального пентафрагмента к той или иной группе антисимметрии (табл.5 описания заявки).
В данном примере: Val - 4, Asp - 4, Cys - 3, Gly - 1, Tyr - 3. Эта числовая последовательность записывается в память программы слева направо 44313 и используется для поиска заданного начального пентафрагмента в папке 0000000000 базы данных в файле 44313_0000000000.
- проведение поиска заданного начального пентафрагмента в базе данных в папке с заданным описанием вторичной структуры пентафрагмента;
Программа обнаружила заданный начальный пентафрагмент в файле 44313_0000000000:
Этот пентафрагмент был выделен из текстового файла, полученного программой PROTEIN 3D на основе обработки координат атомов белка из Protein Data Bank и имеет β-структуру, описываемую как 0000000000, не содержащую Н-связей в ближайшем окружении пентафрагмента.
Таблицы 16, 17, 18 иллюстрируют работу программы. В левой части, озаглавленной «Вввод» размещаются: в первом столбце вводимые в программу PROTCOM порядковые номера проектируемых аминокислот, во втором столбце помещаются аминокислоты при вводе согласно п.З) или пары переменных согласно п.Л), выбираемые оператором на основе заданной вторичной структуры (таблицы 15). В третьем столбце записывается введенное в программу описание вторичной структуры в виде десятизначного числа. В центральной части, озаглавленной «Поиск пентафрагмента в базе данных», помещаются: в первом столбце наименования файлов с номером кодировки и номером заданной папки, а во втором - наименования найденных в пентафрагментах аминокислот. В правой части таблиц проводится запись, осуществляемая программой PROTCOM в рабочем файле после обнаружения заданного начального пентафрагмента, а в дальнейшем - после выбора аминокислоты в файле с номером кодировки номером заданной папки.
- при нахождении в папке заданного начального пентафрагмента считают этот пентафрагмент первым из возможного числа N пентафрагментов проектируемой первичной структуры белка и производят:
- фиксирование номера папки базы данных, содержащей первый пентафрагмент;
- запись последовательности аминокислот первого пентафрагмента в рабочий файл программы;
- запись десятизначного номера папки, описывающего вторичную структуру найденного первого пентафрагмента в рабочий файл;
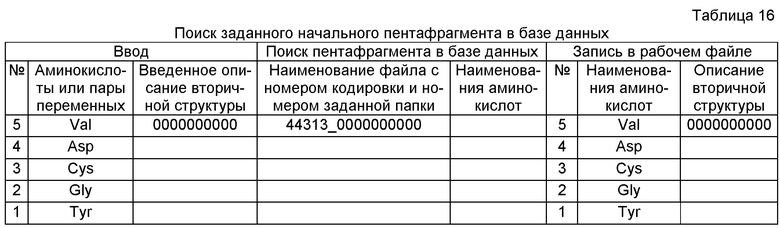
Программа обнаружила введенный начальный пентафрагмент в файле с соответствующей кодировкой и номером папки, и делает запись в рабочем файле (табл.16). Поскольку заданный начальный пентафрагмент был найден, то мы опускаем действия поиска, относящееся к случаю не нахождения в папке заданного начального пентафрагмента.
Л) задают описание вторичной структуры для каждого последующего из (N-1) пентафрагментов, используя описание заданной вторичной структуры в виде последовательности десятизначных булевых чисел, которые соответствуют названиям папок в базе данных, содержащих указанные пентафрагменты, путем введения того же или измененного десятизначного числа, описывающего вторичную структуру предыдущего пентафрагмента, в программу PROTCOM;
Для этого в процессе задания описания вторичной структуры программа PROTCOM предлагает ввести пары переменных 00, 01, 10 или 11. Из таблицы 15 видно, что следующим десятизначным числом является 1000000000. По этой причине оператор выбирает 10, и вводит пару переменных 10 в программу (столбец «Аминокислоты или пары переменных» в таблице 17). Программа добавляет 10 слева и удаляет пару цифр справа, что приводит к изменению десятизначного числа, описывающего вторичную структуру предыдущего пентафрагмента, что отражено в столбце «Описание заданной вторичной структуры» таблицы 17.
М) проводят поиск в базе данных пентафрагментов, содержащих четыре аминокислоты каждого из (N-1) пентафрагментов, записанных в рабочем файле, и одну новую, при этом алгоритм поиска включает в себя:
- выделение и запоминание четырех последних аминокислот в каждом из (N-1) пентафрагментов, записанных в рабочем файле;
- поиск пентафрагментов, содержащих четыре последние аминокислоты каждого из (N-1) пентафрагментов, записанных в рабочем файле, и одну новую аминокислоту в базе данных в папке с заданным описанием вторичной структуры;

Для этого программа выделяет в пентафрагменте, записанном рабочем файле таблицы 16 четыре аминокислоты, записанных сверху вниз: Val, Asp, Cys, Gly.
Далее программа их кодирует в соответствии с принадлежностью к той или иной группе антисимметрии и записывает кодовый номер слева направо, аналогично сформированным индексам файлов, но без первой аминокислоты: 4431 и проводит в базе данных поиск пентафрагментов, содержащих четыре выделенных аминокислоты, в папке с заданной структурой следующего пентафрагмента (1000000000), т.е в файлах Х4431_1000000000, где X может принимать значения 1, 2, 3, 4, соответствующие номерам групп антисимметрии (см. таблицу 5 описания заявки) - 14431_1000000000, 24431_1000000000, 34431_1000000000, 44431_1000000000.
В результате поиска были найдены пентафрагменты, содержащие четыре последних аминокислоты: Val, Asp, Cys, Gly и следующие пятые аминокислоты, записанные вместе с кодами белков, из которых они были получены:
- в файле группы 1 (14431_1000000000): Gly;
- в файле группы 2 (24431_1000000000) - пентафрагменты не найдены;
- в файле группы 3 (34431_1000000000): - пентафрагменты не найдены;
- в файле группы 4 (44431_1000000000): - пентафрагменты не найдены.
Обращаем внимание, что в файлах групп 2, 3, 4 пентафрагменты не найдены. Для проектирования используем единственную аминокислоту Gly.
- при нахождении таких пентафрагментов оператор производит:
- выбор одной из новых аминокислот и присоединение ее к четырем последним аминокислотам предыдущего пентафрагмента;
В качестве пятой аминокислоты оператор выбрал Gly и ввел эту информацию в программу.
Далее программа производит:
- запись новой аминокислоты в рабочий файл («Запись в рабочем файле», табл.17), отражающий проектируемую первичную структуру белка (Gly);
- запись десятичного номера папки, описывающего вторичную структуру каждого найденного пентафрагмента (1000000000);
Поскольку пентафрагмент был найден, то мы опускаем действия поиска, относящееся к случаю не нахождения в папке пентафрагмента.
Далее производят повторение действий согласно пп.Л) и М) до окончания процесса проектирования. Как видно из таблицы 18, в процессе проектирования последовательности аминокислот на всех этапах появлялась возможность выбора лишь из одной аминокислоты.
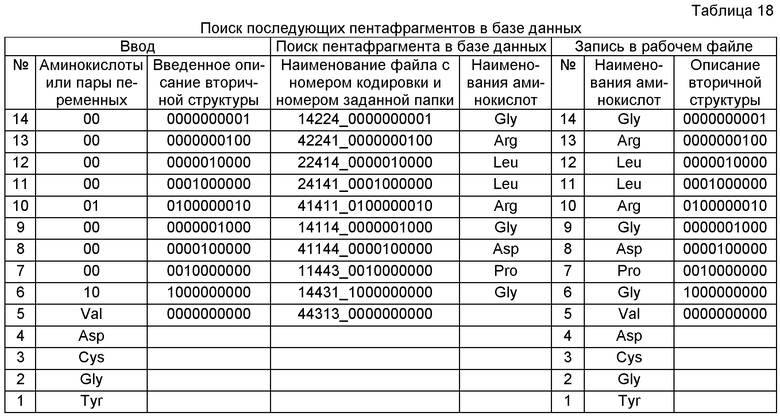
H) спроектированной первичной структурой белка считают полученную в рабочем файле последовательность аминокислот, с соответствующим описанием ее вторичной структуры, записанной в рабочем файле программы PROTCOM и представленная в правой части таблицы 18.
Для экспериментального подтверждения существования спроектированной в примере 2 первичной структуры белка с заданной вторичной структурой в Protein Data Bank было найдено несколько фрагментов белков, имеющих последовательность аминокислот, полностью совпадающую либо со всей спроектированной последовательностью аминокислот примера 2, либо с участком этой последовательности (табл.19).
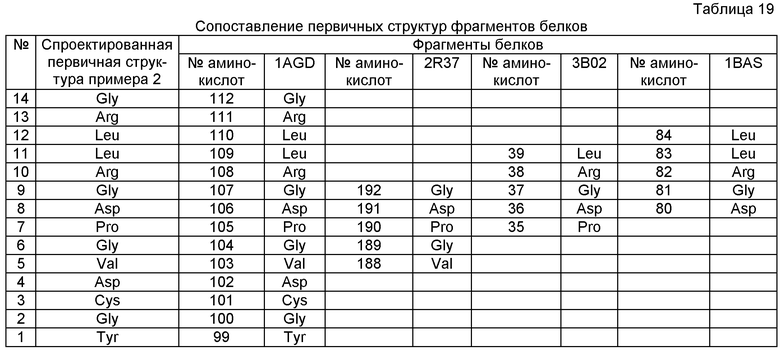
Как следует из таблицы 19, спроектированная нами первичная структура аминокислот для примера 2 оказалась идентична последовательности аминокислот фрагмента белка 1AGD. В таблице 20 приведено двумерное описание водородных связей представленных в таблице 19 фрагментов белков, полученное с помощью программы Protein 3D на основе файлов таблицы 19. Описание их вторичной структуры в виде десятизначных булевых чисел приведено в правом столбце таблицы 20. Для белка 1AGD полностью идентично заданному описанию вторичной структуры спроектированной первичной структуры белка примера 2 (табл.18). Кроме того, найдено, отдельные участки этой последовательности могут быть составлены на основе пентафрагментов белков 2R37 (№9), 3В02 (№11) и 1BAS (№12), которые не имеют никакого родства с белком 1AGD. В таблице 20 приведено описание их вторичной структуры в виде десятизначных булевых чисел, которое полностью совпадает с заданным описанием вторичной структуры спроектированной первичной структуры примера 2.
На фиг.2-4 представлены скринпринты фрагментов вторичной структуры белков 1AGD, 2R37, 3В02, 1BAS, полученные с помощью программы Protein 3D. Сопоставление фрагмента белка 1AGD с фрагментом белка 2R37 (фиг.2,а и 2,б), с фрагментом белка 3В02 (фиг.3,а и 3,б) и фрагментом белка 1BAS (фиг.4,а и 4,б) позволяет сделать вывод, их вторичные структуры идентичны и при этом они взаимозаменяемы. Это означает, что нет разницы в том, спроектирован белок примера 2 на основе пентафрагментов только белка 1AGD или с использованием пентафрагментов, полученных из четырех разных белков - 1AGD, 2R37, 3В02 и 1BAS.
Общий вид исследованного методом PCА белка 1AGD показан на фиг.5,а. В прямоугольнике выделен фрагмент, соответствующий первичной структуре, спроектированный заявляемым способом. На фиг.5,б он показан крупным планом, а на фиг.5,в - детальный вид фрагмента вторичной структуры белка 1GDJ, соответствующий заданной вторичной структуре примера 2. Приведенные фигуры наглядно иллюстрируют присутствие данного фрагмента в реальном белке.
Таким образом, в примере 2 спроектированная первичная структура белка подтверждается двумя вариантами. Первый вариант: первичная структура состоит только из пентафрагментов белка 1AGD. Заданная вторичная структура спроектированного белка примера 2 полностью совпадает с вторичной структурой фрагмента белка 1AGD. Второй вариант: первичная структура состоит из пентафрагментов, полученных из четырех разных белков - 1AGD, 2R37, 3В02 и 1BAS. Описание вторичных структур пентафрагментов белков 1AGD, 2R37, 3В02 и 1BAS также полностью совпадает с заданным описанием вторичной структуры спроектированной первичной структуры белка примера 2.
Сведения о вторичной структуре белков 1AGD, 2R37, 3В02 и 1BAS опубликованы и представлены таблице 21.
Сведения о регистрации базы данных и программы, использованных в описании заявки
«База данных пентафрагментов белков».
Авторы: Карасев В.А., Беляев А.И., Лучинин В.В.
Свидетельство о государственной регистрации базы данных №2010620364
Зарегистрировано в Реестре баз данных 7 июля 2010 г.
«Компьютерная программа для конструирования первичной структуры белка с заданной вторичной структурой» - «PROTCOM».
Авторы: Карасев В.А., Беляев А.И., Лучинин В.В.
Свидетельство о государственной регистрации программы для ЭВМ №2011611105.
Зарегистрировано в Реестре программ для ЭВМ 2 февраля 2011 года.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ПРОГНОЗИРОВАНИЯ ВТОРИЧНОЙ СТРУКТУРЫ БЕЛКА | 2010 |
|
RU2425837C1 |
| КРИСТАЛЛИЧЕСКАЯ СТРУКТУРА ГРЕМЛИН-1 И ИНГИБИРУЮЩЕЕ АНТИТЕЛО | 2017 |
|
RU2769285C2 |
| ПОЛИПЕПТИДЫ, МОДУЛИРУЮЩИЕ SIGLEC-ЗАВИСИМЫЕ ИММУННЫЕ ОТВЕТЫ | 2017 |
|
RU2776807C2 |
| ПЕПТИДНЫЕ АНТАГОНИСТЫ ПЕПТИДНЫХ ГОРМОНОВ ИЗ СЕМЕЙСТВА КАЛЬЦИТОНИНА (CGRP) И ИХ ПРИМЕНЕНИЕ | 2017 |
|
RU2742826C2 |
| ЭКСПРЕССИЯ ПОЛИПЕПТИДОВ НИТРОГЕНАЗЫ В РАСТИТЕЛЬНЫХ КЛЕТКАХ | 2020 |
|
RU2833880C2 |
| РЕКОМБИНАНТНОЕ ПОЛУЧЕНИЕ СТЕВИОЛ-ГЛИКОЗИДОВ | 2014 |
|
RU2741103C2 |
| МУТАНТЫ FIMH E. COLI И ИХ ПРИМЕНЕНИЕ | 2021 |
|
RU2831010C1 |
| CD40L-СПЕЦИФИЧНЫЕ КАРКАСНЫЕ СТРУКТУРЫ, ПРОИСХОДЯЩИЕ ИЗ TN3, И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2012 |
|
RU2826189C2 |
| НОВЫЙ ВАРИАНТ MdtH И СПОСОБ ПОЛУЧЕНИЯ О-ФОСФОСЕРИНА, ЦИСТЕИНА И ПРОИЗВОДНОГО ЦИСТЕИНА С ЕГО ПРИМЕНЕНИЕМ | 2021 |
|
RU2835524C2 |
| МУТИРОВАННЫЙ БЕЛОК SPOT И СПОСОБ ПОЛУЧЕНИЯ L-АМИНОКИСЛОТ С ЕГО ПРИМЕНЕНИЕМ | 2022 |
|
RU2837657C2 |
Изобретение относится к компьютерному способу, использующему биохимические базы данных при разработке новых белковых соединений. Проектирование осуществляется оператором с помощью специально написанной программы PROTCOM на основе использования базы данных пентафрагментов белков. Процесс проектирования состоит в задании и введении в программу PROTCOM начальной последовательности из пяти аминокислот (заданного начального пентафрагмента) и десятизначного числа, записанного в двоичной системе, являющегося описанием вторичной структуры заданного начального пентафрагмента. Проводится поиск этой последовательности в папке базы данных, с номером, соответствующим заданному десятизначному числу. Поиск производят до тех пор, пока заданный начальный пентафрагмент не будет найден в базе данных. После его нахождения считают этот пентафрагмент первым из возможного числа N пентафрагментов проектируемой первичной структуры белка и производят его запись вместе с десятизначным номером папки, описывающим его вторичную структуру, в рабочий файл программы. Далее задают вторичные структуры каждого последующего из (N-1) пентафрагментов путем введения того же или измененного десятизначного числа, описывающего вторичную структуру предыдущего пентафрагмента в программу и проводят поиск в базе данных пентафрагментов, содержащих четыре аминокислоты каждого из (N-1) пентафрагментов, записанных в рабочем файле и одну новую. При нахождении таких пентафрагментов производят выбор одной из новых аминокислот и присоединение ее к четырем последним аминокислотам предыдущего пентафрагмента, запись новой аминокислоты и десятичного номера папки, описывающего вторичную структуру каждого найденного пентафрагмента в рабочий файл. Спроектированной первичной структурой белка считают полученную в рабочем файле последовательность аминокислот, с соответствующим описанием ее вторичной структуры. Предложенный способ проектирования первичной структуры белка существенно упрощает и ускоряет задачу проектирования белков с заданной вторичной структурой. 5 ил., 21 табл., 2 пр.
Способ проектирования первичной структуры белка с заданной вторичной структурой на основе получения характеризующей ее последовательности аминокислот и описания вторичной структуры, заключающийся в следующем:
A) создают базу данных аминокислотных пентафрагментов белков, содержащую папки с пентафрагментами, причем исходный список папок составлен по их названиям, сформированным на основании закодированного в двоичной системе описания водородных связей пептидных групп пентафрагментов во вторичной структуре белков, и записывают ее на информационный носитель;
Б) создают каталог описаний вторичных структур, содержащий описания вторичных структур в виде последовательности 10-значных булевых чисел;
B) вводят в память компьютера записанную на информационный носитель базу данных аминокислотных пентафрагментов белков;
Г) задают описание вторичной структуры проектируемой первичной структуры белка в виде последовательности 10-значных булевых чисел на основе каталога описаний вторичных структур;
Д) задают и вводят в память компьютера начальную последовательность из пяти аминокислот, принадлежащих к группе из двадцати канонических аминокислот белков, являющуюся заданным начальным пентафрагментом;
Е) задают и вводят в память компьютера описание вторичной структуры заданного начального пентафрагмента в виде десятизначного числа в двоичной системе, являющегося первым 10-значным числом в заданном описании вторичной структуры, которое соответствует названию папки в базе данных, содержащей указанный начальный пентафрагмент;
Ж) вводят в память компьютера программу PROTCOM для выделения и поиска пентафрагментов проектируемого белка в базе данных и записи названий аминокислот найденных пентафрагментов и номеров папок базы данных, описывающих вторичную структуру, в которых обнаружены искомые пентафрагменты;
З) вводят и запоминают заданный начальный пентафрагмент проектируемого белка в виде последовательности из пяти аминокислот в программу PROTCOM;
И) вводят и запоминают заданное описание вторичной структуры заданного начального пентафрагмента в виде десятизначного числа в двоичной системе в программу PROTCOM;
К) проводят поиск заданного начального пентафрагмента проектируемого белка в базе данных с помощью ранее записанной в память компьютера программы PROTCOM, при этом алгоритм поиска включает в себя:
- кодирование заданного начального пентафрагмента для целей поиска в базе данных;
- проведение поиска заданного начального пентафрагмента в базе данных в папке с заданным описанием вторичной структуры пентафрагмента;
- при нахождении в папке заданного начального пентафрагмента считают этот пентафрагмент первым из возможного числа N пентафрагментов проектируемой первичной структуры белка и производят:
- фиксирование номера папки базы данных, содержащей первый пентафрагмент;
- запись последовательности аминокислот первого пентафрагмента в рабочий файл программы;
- запись десятизначного номера папки, описывающего вторичную структуру найденного первого пентафрагмента в рабочий файл;
- при не нахождении в папке заданного начального пентафрагмента:
- задают и вводят в память компьютера новую начальную последовательность из пяти аминокислот, принадлежащих к группе из двадцати канонических аминокислот белков, являющуюся новым заданным начальным пентафрагментом;
- вводят и запоминают новый заданный начальный пентафрагмент проектируемого белка в виде последовательности из пяти аминокислот в программу PROTCOM;
- проводят поиск нового заданного начального пентафрагмента проектируемого белка в базе данных с помощью ранее записанной в память компьютера программы PROTCOM, при этом алгоритм поиска включает в себя:
- кодирование нового заданного начального пентафрагмента для целей поиска в базе данных;
- проведение поиска нового заданного начального пентафрагмента в базе данных в папке с заданной вторичной структурой пентафрагмента;
- повторение задания новых начальных пентафрагментов и поиска новых заданных исходных пентафрагментов осуществляют до тех пор, пока не будет найден пентафрагмент с такой последовательностью аминокислот, которая находится в папке базы данных, описывающей заданную вторичную структуру пентафрагмента;
Л) задают описание вторичной структуры для каждого последующего из (N-1) пентафрагментов, используя описание заданной вторичной структуры в виде последовательности 10-значных булевых чисел, которые соответствуют названиям папок в базе данных, содержащих указанные пентафрагменты, путем введения того же или измененного десятизначного числа, описывающего вторичную структуру предыдущего пентафрагмента, в программу PROTCOM;
М) проводят поиск в базе данных пентафрагментов, содержащих четыре аминокислоты каждого из (N-1) пентафрагментов, записанных в рабочем файле и одну новую, при этом алгоритм поиска включает в себя:
- выделение и запоминание четырех последних аминокислот в каждом из (N-1) пентафрагментов, записанных в рабочем файле;
- поиск пентафрагментов, содержащих четыре последние аминокислоты каждого из (N-1) пентафрагментов, записанных в рабочем файле, и одну новую аминокислоту в базе данных в папке с заданным описанием вторичной структуры;
- при нахождении таких пентафрагментов производят:
- выбор одной из новых аминокислот и присоединение ее к четырем последним аминокислотам предыдущего пентафрагмента;
- запись новой аминокислоты в рабочий файл, отражающий проектируемую первичную структуру белка;
- запись десятичного номера папки, описывающего вторичную структуру каждого найденного пентафрагмента;
- при не нахождении таких пентафрагментов производят:
- задание измененного описания вторичной структуры;
- выделение четырех последних аминокислот в последующем пентафрагменте;
- поиск пентафрагментов, содержащих четыре последних аминокислоты предыдущего пентафрагмента и одну новую аминокислоту в базе данных в папке с измененным описанием вторичной структуры;
- повторение изменения описания вторичной структуры и поиск в базе данных осуществляют до тех пор, пока не будет найден хотя бы один пентафрагмент, содержащий четыре аминокислоты предыдущего пентафрагмента;
Н) спроектированной первичной структурой белка считают полученную в рабочем файле последовательность аминокислот, с соответствующим описанием ее вторичной структуры.
| LIANG DAI et al | |||
| «Improving computational protein design by using structure derived sequence profile», Proteins, 2010 August 1, 78(10), 2338-2348 | |||
| YI LIU et al | |||
| «RosettaDesign server for protein design», Nucleic Acids Res | |||
| Пломбировальные щипцы | 1923 |
|
SU2006A1 |
| XAVIER I et al | |||
| «Computational | |||
Авторы
Даты
2014-04-10—Публикация
2011-09-22—Подача