Перекрестная ссылка на родственные заявки
В настоящей заявке испрашивается преимущество приоритета предварительной заявки США 61/134094, поданной 2 июля 2008 г., предварительной заявки США 61/133947, поданной 2 июля 2008 г., и предварительной заявки США 61/134011, поданной 2 июля 2008 г., содержание которых во всей своей полноте вводится в настоящее описание посредством ссылки.
Предшествующий уровень техники
Изопрен (2-метил-1,3-бутадиен) является ценным исходным материалом, используемым для получения различных синтетических полимеров, а чаще всего, для производства синтетических каучуков. В природе изопрен продуцируется различными микробами, растениями и животными. В частности, было выявлено два пути биосинтеза изопрена: мевалонатный путь (MVA) и не-мевалонатный путь (DXP) (фигура 19). Однако, с коммерческой точки зрения, выход изопрена, продуцируемого природными микроорганизмами, является недостаточным. Примерно 800000 тонн цис-полиизопрена в год продуцируется в результате полимеризации изопрена; причем, в большинстве случаев, этот полиизопрен применяется в производстве шин и в резиновой промышленности. Изопрен также подвергают сополимеризации для его использования в качестве синтетического эластомера в других продуктах, таких как обувь, механические изделия, медицинские препараты, спортивные товары и латекс.
В настоящее время, производство шин и резиновая промышленность базируются на применении природного и синтетического каучука. Природный каучук получают из млечного сока каучуковых деревьев, произрастающих в дождевых лесах Африки. Синтетический каучук получают, главным образом, на основе бутадиеновых полимеров. Для производства таких полимеров, бутадиен получают промышленным способом в виде продукта сополимеризации этилена и пропилена.
Изопрен может быть получен путем ректификации нефти, однако очистка этого материала является дорогостоящей и требует много времени. При нефтяном крекинге потока C5-углеводородов продуцируется примерно лишь 15% изопрена. А поэтому необходимо разработать более экономичный метод получения изопрена. В частности, желательно разработать методы продуцирования изопрена, которые обеспечивали бы скорость продуцирования, а также титры и частоту изопрена, отвечающие современным требованиям экономически выгодного промышленного производства. Было бы также желательно создать системы для продуцирования изопрена, которые состояли бы из недорогостоящих исходных материалов.
Описание сущности изобретения
В одном из своих аспектов, настоящее изобретение относится к клеточным культурам, продуцирующим изопрен. В некоторых своих вариантах, настоящее изобретение относится к культуре клеток, которая продуцирует более чем примерно 400 нмоль изопрена/грамм клеток в расчете на сырую массу клеток/час (нмоль/гwcm/час). В некоторых вариантах изобретения, клетки имеют гетерологичную нуклеиновую кислоту, которая (i) кодирует полипептид изопренсинтазы и (ii) функционально присоединена к промотору. В некоторых вариантах изобретения, клетки культивируют в культуральной среде, которая включает источник углерода, такой как, но не ограничивающийся им, углевод, глицерол, глицерин, дигидроксиацетон, источник, содержащий один атом углерода, масло, животный жир, масло животного происхождения, жирная кислота, липид, фосфолипид, глицеролипид, моноглицерид, диглицерид, триглицерид, возобновляемый источник углерода, полипептид (например, белок или пептид микробного или растительного происхождения), дрожжевой экстракт, компонент дрожжевого экстракта или любая комбинация двух или более вышеуказанных соединений. В некоторых вариантах изобретения, клетки культивируют в условиях с ограниченным содержанием глюкозы.
В некоторых своих вариантах, настоящее изобретение относится к клеткам, которые, при их культивировании в среде для культивирования клеток, обладают способностью превращать примерно более чем 0,002% углерода в изопрен. В некоторых вариантах изобретения, клетки имеют гетерологичную нуклеиновую кислоту, которая (i) кодирует полипептид изопренсинтазы и (ii) функционально присоединена к промотору. В некоторых вариантах изобретения, клетки культивируют в культуральной среде, которая включает источник углерода, такой как, но не ограничивающийся им, углевод, глицерол, глицерин, дигидроксиацетон, источник, содержащий один атом углерода, масло, животный жир, масло животного происхождения, жирная кислота, липид, фосфолипид, глицеролипид, моноглицерид, диглицерид, триглицерид, возобновляемый источник углерода, полипептид (например, белок или пептид микробного или растительного происхождения), дрожжевой экстракт, компонент дрожжевого экстракта или любая комбинация двух или более вышеуказанных соединений. В некоторых вариантах изобретения, клетки культивируют в условиях с ограниченным содержанием глюкозы.
В некоторых своих вариантах, настоящее изобретение относится к клеточной культуре, которая содержит гетерологичную нуклеиновую кислоту, кодирующую полипептид изопренсинтазы. В некоторых вариантах изобретения, клетки имеют гетерологичную нуклеиновую кислоту, которая (i) кодирует полипептид изопренсинтазы и (ii) функционально присоединена к промотору. В некоторых вариантах изобретения, клетки культивируют в культуральной среде, которая включает источник углерода, такой как, но не ограничивающийся им, углевод, глицерол, глицерин, дигидроксиацетон, источник, содержащий один атом углерода, масло, животный жир, масло животного происхождения, жирная кислота, липид, фосфолипид, глицеролипид, моноглицерид, диглицерид, триглицерид, возобновляемый источник углерода, полипептид (например, белок или пептид микробного или растительного происхождения), дрожжевой экстракт, компонент дрожжевого экстракта или любая комбинация двух или более вышеуказанных соединений. В некоторых вариантах изобретения, клетки культивируют в условиях с ограниченным содержанием глюкозы.
В одном из своих аспектов, настоящее изобретение относится к способам продуцирования изопрена, таким как способы, осуществляемые с использованием любых описанных здесь клеток, продуцирующих изопрен. В некоторых вариантах изобретения, указанный способ включает культивирование клеток в условиях, достаточных для продуцирования более чем примерно 400 нмоль/гwcm/час изопрена. В некоторых вариантах изобретения, указанный способ также включает регенерацию изопрена, продуцированного данными клетками. В некоторых вариантах изобретения, указанный способ включает очистку изопрена, продуцированного данными клетками. В некоторых вариантах изобретения, указанный способ включает полимеризацию изопрена. В некоторых вариантах изобретения, указанные клетки имеют гетерологичную нуклеиновую кислоту, которая (i) кодирует полипептид изопренсинтазы и (ii) функционально присоединена к промотору. В некоторых вариантах изобретения, клетки культивируют в культуральной среде, которая включает источник углерода, такой как, но не ограничивающийся ими, углевод, глицерол, глицерин, дигидроксиацетон, источник, содержащий один атом углерода, масло, животный жир, масло животного происхождения, жирная кислота, липид, фосфолипид, глицеролипид, моноглицерид, диглицерид, триглицерид, возобновляемый источник углерода, полипептид (например, белок или пептид микробного или растительного происхождения), дрожжевой экстракт, компонент дрожжевого экстракта или любая комбинация двух или более вышеуказанных соединений. В некоторых вариантах изобретения, клетки культивируют в условиях с ограниченным содержанием глюкозы. В различных вариантах изобретения, количество изопрена (такое как общее количество продуцируемого изопрена или количество изопрена, продуцируемое на литр бульона в час на OD600), продуцируемое в стационарной фазе, более чем или примерно в 2 или более раз превышает количество изопрена, продуцируемого в фазе роста в течение того же самого периода времени. В некоторых вариантах изобретения, газовая фаза содержит более чем или примерно 9,5% (по объему) кислорода, а концентрация изопрена в этой газовой фазе меньше нижнего предела воспламеняемости или выше верхнего предела воспламеняемости. В конкретных вариантах изобретения, (i) концентрация изопрена в газовой фазе меньше нижнего предела воспламеняемости или выше верхнего предела воспламеняемости, и (ii) указанные клетки продуцируют более чем примерно 400 нмоль/гwcm/час изопрена.
В некоторых вариантах изобретения, указанный способ включает культивирование клеток в условиях, достаточных для превращения более чем примерно 0,002% углерода (моль/моль) в среде для культивирования клеток в изопрен. В некоторых вариантах изобретения, указанный способ также включает регенерацию изопрена, продуцированного данными клетками. В некоторых вариантах изобретения, указанный способ включает очистку изопрена, продуцированного данными клетками. В некоторых вариантах изобретения, указанный способ включает полимеризацию изопрена. В некоторых вариантах изобретения, указанные клетки имеют гетерологичную нуклеиновую кислоту, которая (i) кодирует полипептид изопренсинтазы и (ii) функционально присоединена к промотору. В некоторых вариантах изобретения, клетки культивируют в культуральной среде, которая включает источник углерода, такой как, но не ограничивающийся им, углевод, глицерол, глицерин, дигидроксиацетон, источник, содержащий один атом углерода, масло, животный жир, масло животного происхождения, жирная кислота, липид, фосфолипид, глицеролипид, моноглицерид, диглицерид, триглицерид, возобновляемый источник углерода, полипептид (например, белок или пептид микробного или растительного происхождения), дрожжевой экстракт, компонент дрожжевого экстракта или любая комбинация двух или более вышеуказанных соединений. В некоторых вариантах изобретения, клетки культивируют в условиях с ограниченным содержанием глюкозы.
В некоторых вариантах изобретения, изопрен продуцируется лишь в стационарной фазе. В некоторых вариантах изобретения, изопрен продуцируется в фазе роста и в стационарной фазе. В различных вариантах изобретения, количество изопрена (такое как общее количество продуцируемого изопрена или количество изопрена, продуцируемое на литр бульона в час на OD600), продуцируемое в стационарной фазе, более чем или примерно в 2, 3, 4, 5, 10, 20, 30, 40, 50 или более раз превышает количество изопрена, продуцируемое в фазе роста в течение того же самого периода времени.
В одном из своих аспектов, настоящее изобретение относится к композициям и системам, содержащим изопрен. В некоторых вариантах изобретения, указанная композиция включает более чем или примерно 2, 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 мг изопрена. В некоторых вариантах изобретения, указанная композиция включает более чем или примерно 2, 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100 г изопрена по массе летучей органической фракции данной композиции.
В некоторых вариантах изобретения, указанная композиция включает более чем или примерно 99,90; 99,92; 99,94; 99,96; 99,98 или 100% изопрена по общей массе всех C5-углеводородов в данной композиции. В некоторых вариантах изобретения, указанная композиция включает менее чем или примерно 0,12, 0,10, 0,08, 0,06, 0,04, 0,02, 0,01, 0,005, 0,001, 0,0005, 0,0001, 0,00005 или 0,00001% C5-углеводородов, не являющихся изопреном (таких как 1,3-циклопентадиен, цис-1,3-пентадиен, транс-1,3-пентадиен, 1-пентин, 2-пентин, 1-пентен, 2-метил-1-бутен, 3-метил-1-бутин, транс-пиперилен, цис-пиперилен, пент-4-ен-1-ин, транс-пент-3-ен-1-ин или цис-пент-3-ен-1-ин), по общей массе всех C5-углеводородов в указанной композиции. В некоторых вариантах изобретения, указанная композиция содержит менее чем или примерно 0,12, 0,10, 0,08, 0,06, 0,04, 0,02, 0,01, 0,005, 0,001, 0,0005, 0,0001, 0,00005 или 0,00001% 1,3-циклопентадиена, цис-1,3-пентадиена, транс-1,3-пентадиена, 1-пентина, 2-пентина, 1-пентена, 2-метил-1-бутена, 3-метил-1-бутина, транс-пиперилена, цис-пиперилена, пент-4-ен-1-ина, транс-пент-3-ен-1-ина или цис-пент-3-ен-1-ина по общей массе всех C5-углеводородов в указанной композиции. В конкретных вариантах изобретения, указанная композиция содержит более чем примерно 2 мг изопрена и более чем или примерно 99,90, 99,92, 99,94, 99,96, 99,98 или 100% изопрена по общей массе всех C5-углеводородов в указанной композиции.
В некоторых вариантах изобретения, указанная композиция содержит менее чем или примерно 50, 40, 30, 20, 10, 5, 1, 0,5, 0,1, 0,05, 0,01 или 0,005 мкг/л любого соединения, которое ингибирует полимеризацию изопрена. В конкретных вариантах изобретения, указанная композиция также содержит более чем примерно 2 мг изопрена.
В некоторых вариантах изобретения, указанная композиция содержит одно или более соединений, выбранных из группы, состоящей из этанола, ацетона, прениловых C5-спиртов и изопреноидных соединений, содержащих 10 или более атомов углерода. В некоторых вариантах изобретения, указанная композиция содержит более чем или примерно 0,005, 0,01, 0,05, 0,1, 0,5, 1, 5, 10, 20, 30, 40, 60, 80, 100 или 120 мкг/л этанола, ацетона, пренилового C5-спирта (такого как 3-метил-3-бутен-1-ол или 3-метил-2-бутен-1-ол), или любых двух или более вышеуказанных соединений. В конкретных вариантах изобретения, указанная композиция содержит более чем примерно 2 мг изопрена и одно или более соединений, выбранных из группы, состоящей из этанола, ацетона, прениловых C5-спиртов и изопреноидных соединений, содержащих 10 или более атомов углерода.
В некоторых вариантах изобретения, указанная композиция включает изопрен и одно или более вторых соединений, выбранных из группы, состоящей из 2-гептанона, 6-метил-5-гептен-2-она, 2,4,5-триметилпиридина, 2,3,5-триметилпиразина, цитронеллаля, ацетальдегида, метантиола, метилацетата, 1-пропанола, диацетила, 2-бутанона, 2-метил-3-бутен-2-ола, этилацетата, 2-метил-1-пропанола, 3-метил-1-бутаналя, 3-метил-2-бутанона, 1-бутанола, 2-пентанона, 3-метил-1-бутанола, этилизобутирата, 3-метил-2-бутенaла, бутилацетата, 3-метилбутилацетата, 3-метил-3-бут-1-енилацетата, 3-метил-2-бут-1-енилацетата, (E)-3,7-диметил-1,3,6-октатриена, (Z)-3,7-диметил-1,3,6-октатриена и 2,3-циклогептенолпиридина. В различных вариантах изобретения, количество одного из этих вторых компонентов по отношению к количеству изопрена, выраженное в процентах по массе (то есть масса компонента, деленная на массу изопрена × 100), превышает или составляет примерно 0,01, 0,02, 0,05, 0,1, 0,5, 1, 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100 или 110% (масс/масс).
В некоторых вариантах изобретения, указанная композиция включает (i) газовую фазу, содержащую изопрен и (ii) клеточную культуру, продуцирующую более чем примерно 400 нмоль/гwcm/час изопрена. В некоторых вариантах изобретения, указанная композиция включает закрытую систему, а газовая фаза включает более чем или примерно 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100 мкг/л изопрена, нормализованные на 1 мл культуры при OD600=1, культивированной в течение 1 часа. В некоторых вариантах изобретения, указанная композиция включает открытую систему, а газовая фаза включает более чем или примерно 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100 мкг/л изопрена, барботированного при скорости 1 vvm (объем воздуха/объем бульона/минуту). В некоторых вариантах изобретения, летучая органическая фракция газовой фазы содержит более чем или примерно 99,90; 99,92; 99,94; 99,96; 99,98 или 100% изопрена по общей массе всех C5-углеводородов в данной летучей органической фракции. В некоторых вариантах изобретения, летучая органическая фракция газовой фазы содержит менее чем или примерно 0,12, 0,10, 0,08, 0,06, 0,04, 0,02, 0,01, 0,005, 0,001, 0,0005, 0,0001, 0,00005 или 0,00001% С5-углеводородов, не являющихся изопреном (таких как 1,3-циклопентадиен, цис-1,3-пентадиен, транс-1,3-пентадиен, 1-пентин, 2-пентин, 1-пентен, 2-метил-1-бутен, 3-метил-1-бутин, транс-пиперилен, цис-пиперилен, пент-4-ен-1-ин, транс-пент-3-ен-1-ин или цис-пент-3-ен-1-ин), по общей массе всех C5-углеводородов в указанной летучей органической фракции. В некоторых вариантах изобретения, летучая органическая фракция газовой фазы содержит менее чем или примерно 0,12, 0,10, 0,08, 0,06, 0,04, 0,02, 0,01, 0,005, 0,001, 0,0005, 0,0001, 0,00005 или 0,00001% 1,3-циклопентадиена, цис-1,3-пентадиена, транс-1,3-пентадиена, 1-пентина, 2-пентина, 1-пентена, 2-метил-1-бутена, 3-метил-1-бутина, транс-пиперилена, цис-пиперилена, пент-4-ен-1-ина, транс-пент-3-ен-1-ина или цис-пент-3-ен-1-ина по общей массе всех C5-углеводородов в указанной летучей органической фракции. В конкретных вариантах изобретения, указанная летучая органическая фракция газовой фазы содержит более чем примерно 2 мг изопрена и более чем или примерно 99,90, 99,92, 99,94, 99,96, 99,98 или 100% изопрена по общей массе всех C5-углеводородов в указанной летучей органической фракции.
В некоторых вариантах изобретения, указанная летучая органическая фракция газовой фазы содержит менее чем или примерно 50, 40, 30, 20, 10, 5, 1, 0,5, 0,1, 0,05, 0,01 или 0,005 мкг/л любого соединения в летучей органической фракции газовой фазы, которое ингибирует полимеризацию изопрена. В конкретных вариантах изобретения, указанная летучая органическая фракция газовой фазы также содержит более чем примерно 2 мг изопрена.
В некоторых вариантах изобретения, указанная летучая органическая фракция газовой фазы содержит одно или более соединений, выбранных из группы, состоящей из этанола, ацетона, прениловых C5-спиртов и изопреноидных соединений, содержащих 10 или более атомов углерода. В некоторых вариантах изобретения, указанная летучая органическая фракция газовой фазы содержит более чем или примерно 0,005, 0,01, 0,05, 0,1, 0,5, 1, 5, 10, 20, 30, 40, 60, 80, 100 или 120 мкг/л этанола, ацетона, пренилового C5-спирта (такого как 3-метил-3-бутен-1-ол или 3-метил-2-бутен-1-ол), или любых двух или более вышеуказанных соединений. В конкретных вариантах изобретения, указанная летучая органическая фракция газовой фазы содержит более чем примерно 2 мг изопрена и одно или более соединений, выбранных из группы, состоящей из этанола, ацетона, прениловых C5-спиртов и изопреноидных соединений, содержащих 10 или более атомов углерода.
В некоторых вариантах изобретения, указанная летучая органическая фракция газовой фазы включает изопрен и одно или более вторых соединений, выбранных из группы, состоящей из 2-гептанона, 6-метил-5-гептен-2-она, 2,4,5-триметилпиридина, 2,3,5-триметилпиразина, цитронеллаля, ацетальдегида, метантиола, метилацетата, 1-пропанола, диацетила, 2-бутанона, 2-метил-3-бутен-2-ола, этилацетата, 2-метил-1-пропанола, 3-метил-1-бутаналя, 3-метил-2-бутанона, 1-бутанола, 2-пентанона, 3-метил-1-бутанола, этилизобутирата, 3-метил-2-бутенaла, бутилацетата, 3-метилбутилацетата, 3-метил-3-бут-1-енилацетата, 3-метил-2-бут-1-енилацетата, (E)-3,7-диметил-1,3,6-октатриена, (Z)-3,7-диметил-1,3,6-октатриена и 2,3-циклогептенолпиридина. В различных вариантах изобретения, количество одного из этих вторых компонентов по отношению к количеству изопрена, выраженное в процентах по массе (то есть масса компонента, деленная на массу изопрена × 100), превышает или составляет примерно 0,01, 0,02, 0,05, 0,1, 0,5, 1, 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, или 110% (масс/масс) в указанной летучей органической фракции газовой фазы.
В некоторых вариантах любой из композиций согласно изобретению, по меньшей мере часть изопрена присутствует в газовой фазе. В некоторых вариантах изобретения, по меньшей мере часть изопрена присутствует в жидкой фазе (такой как конденсат). В некоторых вариантах изобретения, по меньшей мере часть изопрена присутствует в твердой фазе. В некоторых вариантах изобретения, по меньшей мере часть изопрена адсорбируется на твердом носителе, таком как носитель, который включает двуокись кремния и/или активированный уголь. В некоторых вариантах изобретения, указанная композиция включает этанол. В некоторых вариантах изобретения, указанная композиция включает примерно от 75 до 90 масс.% этанола, например, примерно от 75 до 80%, примерно от 80 до 85%, или примерно от 85 до 90% масс.% этанола. В некоторых вариантах изобретения, указанная композиция включает примерно от 4 до 15 масс.% изопрена, например, примерно от 4 до 8%, примерно от 8 до 12%, или примерно от 12 до 15% масс.% изопрена.
В некоторых своих вариантах, настоящее изобретение относится к системам, которые включают любые клетки и/или композиции, описанные в настоящей заявке. В некоторых вариантах изобретения, указанная система также включает реактор, камера которого содержит клеточную культуру, продуцирующую более чем примерно 400, 500, 600, 700, 800, 900, 1000, 1250, 1500, 1750, 2000, 2500, 3000, 4000, 5000 или более нмоль/гwcm/час изопрена. В некоторых вариантах изобретения, указанная система не является закрытой системой. В некоторых вариантах изобретения, по меньшей мере часть изопрена удалена из этой системы. В некоторых вариантах изобретения, указанная система включает газовую фазу, содержащую изопрен. В различных вариантах изобретения, указанная газовая фаза содержит любую из описанных здесь композиций.
В одном из своих аспектов, настоящее изобретение относится к шинам, содержащим полиизопрен. В некоторых вариантах изобретения, полиизопрен получают путем (i) полимеризации изопрена в любой из описанных здесь композиций или (ii) полимеризации изопрена, регенерированного из любых описанных здесь композиций. В некоторых вариантах изобретения, полиизопрен включает цис-1,4-полиизопрен.
В некоторых вариантах любых композиций, систем и способов согласно изобретению изопрен продуцируется в газовой фазе в апирогенных концентрациях. В некоторых вариантах изобретения, газовая фаза содержит примерно менее чем 9,5% (по объему) кислорода. В некоторых вариантах изобретения, газовая фаза содержит более чем или примерно 9,5% (по объему) кислорода, а концентрация изопрена в газовой фазе ниже нижнего предела воспламеняемости или выше верхнего предела воспламеняемости. В некоторых вариантах изобретения, часть газовой фазы, не являющейся изопреном, содержит примерно от 0% до 100% (по объему) кислорода, например, примерно от 10% до 100% (по объему) кислорода. В некоторых вариантах изобретения, часть газовой фазы, не являющейся изопреном, содержит примерно от 0% до 99% (по объему) азота. В некоторых вариантах изобретения, часть газовой фазы, не являющейся изопреном, содержит примерно от 1% до 50% (по объему) CO2.
В некоторых вариантах любого из аспектов изобретения, клеточные культуры продуцируют изопрен в количествах, превышающих или примерно составляющих 400 500, 600, 700, 800, 900, 1000, 1250, 1500, 1750, 2000, 2500, 3000, 4000, 5000 или более нмоль/гwcm/час изопрена. В некоторых вариантах любого из аспектов изобретения, клеточные культуры превращают более чем или примерно 0,002, 0,005, 0,01, 0,02, 0,05, 0,1, 0,12, 0,14, 0,16, 0,2, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9, 1,0, 1,2, 1,4, 1,6% или более углерода в клеточной культуральной среде в изопрен. В некоторых вариантах любого из аспектов изобретения, клеточные культуры продуцируют изопрен в количествах, превышающих или примерно составляющих 1, 10, 25, 50, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900, 1000, 1250, 1500, 1750, 2000, 2500, 3000, 4000, 5000, 10000, 100000 или более нг изопрена/грамм сырой массы клеток/час (нг/гwcm/ч). В некоторых вариантах любого из аспектов изобретения, клеточные культуры продуцируют кумулятивный титр (общее количество) изопрена, превышающий или составляющий примерно 1, 10, 25, 50, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900, 1000, 1250, 1500, 1750, 2000, 2500, 3000, 4000, 5000, 10000, 50000, 100000 или более мг изопрена/л бульона (мг/лбульона, где объем бульона включает объем клеток и клеточной среды). Другие репрезентативные уровни продуцирования изопрена и общее количество продуцируемого изопрена описаны в настоящей заявке.
В некоторых вариантах любого из аспектов изобретения, клетки также содержат гетерологичную нуклеиновую кислоту, кодирующую полипептид IDI. В некоторых вариантах любого из аспектов изобретения, клетки также содержат встроенную копию эндогенной нуклеиновой кислоты, кодирующей полипептид IDI. В некоторых вариантах любого из аспектов изобретения, клетки также содержат гетерологичную нуклеиновую кислоту, кодирующую полипептид DXS. В некоторых вариантах любого из аспектов изобретения, клетки также содержат встроенную копию эндогенной нуклеиновой кислоты, кодирующей полипептид DXS. В некоторых вариантах любого из аспектов изобретения, клетки также содержат одну или более нуклеиновых кислот, кодирующих полипептид IDI и полипептид DXS. В некоторых вариантах любого из аспектов изобретения, одна нуклеиновая кислота кодирует полипептид изопренсинтазы, полипептид IDI и полипептид DXS. В некоторых вариантах любого из аспектов изобретения, один вектор кодирует полипептид изопренсинтазы, полипептид IDI и полипептид DXS. В некоторых вариантах изобретения, указанный вектор содержит селективный маркер, такой как нуклеиновая кислота, резистентная к антибиотикам.
В некоторых вариантах любого из аспектов изобретения, гетерологичная нуклеиновая кислота изопренсинтазы функционально присоединена к промотору T7, такому как промотор T7, содержащийся в среднекопийной или высококопийной плазмиде. В некоторых вариантах любого из аспектов изобретения, гетерологичная нуклеиновая кислота изопренсинтазы функционально присоединена к промотору Trc, такому как промотор Trc, содержащийся в среднекопийной или высококопийной плазмиде. В некоторых вариантах любого из аспектов изобретения, гетерологичная нуклеиновая кислота изопренсинтазы функционально присоединена к промотору Lac, такому как промотор Lac, содержащийся в низкокопийной плазмиде. В некоторых вариантах любого из аспектов изобретения, гетерологичная нуклеиновая кислота изопренсинтазы функционально присоединена к эндогенному промотору, такому как эндогенный промотор щелочной сериновой протеазы. В некоторых вариантах изобретения, гетерологичная нуклеиновая кислота изопренсинтазы интегрируется в хромосому клеток без селективного маркера.
В некоторых вариантах изобретения, одна или более нуклеиновых кислот пути MVA, а также нуклеиновых кислот IDI, DXP или изопренсинтазы находятся под контролем промотора или фактора, который является более активным в стационарной фазе, чем в фазе роста. Так, например, одна или более нуклеиновых кислот пути MVA, а также нуклеиновых кислот IDI, DXP или изопренсинтазы могут находиться под контролем фактора сигма в стационарной фазе, такого как RpoS. В некоторых вариантах изобретения, одна или более нуклеиновых кислот пути MVA, а также нуклеиновых кислот IDI, DXP или изопренсинтазы находятся под контролем промотора, который является индуцибельным в стационарной фазе, такого как промотор, индуцируемый регулятором ответа, который является активным в стационарной фазе.
В некоторых вариантах любого из аспектов изобретения, по меньшей мере часть клеток сохраняет гетерологичную нуклеиновую кислоту изопренсинтазы в процессах по меньшей мере или примерно 5, 10, 20, 40, 50, 60, 65 или более делений клеток в непрерывной культуре (такой как непрерывная культура без разведения). В некоторых вариантах любого из аспектов изобретения, нуклеиновая кислота, содержащая нуклеотидную последовательность изопренсинтазы, IDI или DXS, также включает селективный маркер, такой как нуклеиновая кислота, резистентная к антибиотикам.
В некоторых вариантах любого из аспектов изобретения, клетки также содержат гетерологичную нуклеиновую кислоту, кодирующую полипептид пути MVA (такой как полипептид пути MVA от Saccharomyces cerevisia или Enterococcus faecalis). В некоторых вариантах любого из аспектов изобретения, клетки содержат встроенную копию эндогенной нуклеиновой кислоты, кодирующей полипептид пути MVA (такой как полипептид пути MVA от Saccharomyces cerevisia или Enterococcus faecalis). В некоторых вариантах любого из аспектов изобретения, клетки также содержат нуклеиновую кислоту изопренсинтазы, DXS и пути MVA. В некоторых вариантах любого из аспектов изобретения, клетки содержат нуклеиновую кислоту изопренсинтазы, нуклеиновую кислоту DXS, нуклеиновую кислоту IDI и нуклеиновую кислоту пути MVA (в добавление к нуклеиновой кислоте IDI).
В некоторых вариантах любого из аспектов изобретения, полипептидом изопренсинтазы является природный полипептид, выделенный из растения, такого как пуэрария (например, Pueraria montana или Pueraria lobata).
В некоторых вариантах любого из аспектов изобретения, указанными клетками являются бактериальные клетки, такие как грам-положительные бактериальные клетки (например, клетки Bacillus, такие как клетки Bacillus subtilis или клетки Streptomyces, такие как клетки Streptomyces lividans, Streptomyces coelicolor или Streptomyces griseus). В некоторых вариантах любого из аспектов изобретения, указанными клетками являются грам-отрицательные бактериальные клетки, (например, клетки Escherichia, такие как клетки Escherichia coli или Pantoea, такие как Pantoea citrea). В некоторых вариантах любого из аспектов изобретения, указанными клетками являются клетки грибов, такие как клетки нитчатых грибов (например, клетки Trichoderma, такие как клетки Trichoderma reesei или клетки Aspergillus, такие как Aspergillus oryzae и Aspergillus niger) или дрожжевые клетки (например, клетки Yarrowia, такие как клетки Yarrowia lipolytica).
В некоторых вариантах любого из аспектов изобретения, источник углерода микробного полипептида включает один или более полипептидов, выделенных из дрожжей или бактерий. В некоторых вариантах любого из аспектов изобретения, источник углерода растительного полипептида включает один или более полипептидов, выделенных из сои, кукурузы, канолы, ятропы, пальмы, арахиса, подсолнечника, кокосового ореха, горчицы, рапса, семян хлопчатника, косточек плодов пальмы, маслин, сафлора, кунжута или семян льна.
В одном из своих аспектов, настоящее изобретение относится к продукту, продуцируемому с использованием любой из композиций или с применением любых способов согласно изобретению.
Краткое описание графического материала
На фигуре 1 представлена нуклеотидная последовательность гена изопренсинтазы пуэрарии, оптимизированная по кодонам для экспрессии в E.coli (SEQ ID NO:1). Старт-кодон показан курсивом, стоп-кодон выделен жирным шрифтом, а добавленный PstI-сайт подчеркнут.
На фигуре 2 представлена карта pTrcKudzu.
На фигуре 3 представлена нуклеотидная последовательность pTrcKudzu (SEQ ID NO:2). Сайт связывания с рибосомой (RBS) подчеркнут, старт-кодон изопренсинтазы пуэрарии показан заглавными буквами жирным шрифтом, а стоп-кодон показан заглавными буквами, выделенными жирным курсивом. Остовом этого вектора является pTrcHis2B.
На фигуре 4 представлена карта pETNHisKudzu.
На фигуре 5 представлена нуклеотидная последовательность pETNHisKudzu (SEQ ID NO:5).
На фигуре 6 представлена карта pCL-lac-Kudzu.
На фигуре 7 представлена нуклеотидная последовательность pCL-lac-Kudzu (SEQ ID NO:7).
На фигуре 8А представлен график, иллюстрирующий продуцирование изопрена в клетках E.coli BL21, не содержащих вектора.
На фигуре 8В представлен график, иллюстрирующий продуцирование изопрена в клетках E.coli BL21, содержащих вектор pCL-lac-Kudzu.
На фигуре 8С представлен график, иллюстрирующий продуцирование изопрена в клетках E.coli BL21, содержащих вектор pTrcKudzu.
На фигуре 8D представлен график, иллюстрирующий продуцирование изопрена в клетках E.coli BL21, содержащих вектор pETN-HisKudzu.
На фигуре 9А представлен график, иллюстрирующий OD в процессе ферментации E.coli BL21/pTrcKudzu в 14-литровом ферментере с периодической культурой с подпиткой.
На фигуре 9В представлен график, иллюстрирующий продуцирование изопрена в процессе ферментации E.coli BL21/pTrcKudzu в 14-литровом ферментере с периодической культурой с подпиткой.
На фигуре 10A представлен график, иллюстрирующий продуцирование изопрена в Panteoa citrea. Контрольные клетки не содержали рекомбинантной изопренсинтазы пуэрарии. Серые ромбы представляют синтез изопрена, а черные квадраты представляют OD600.
На фигуре 10B представлен график, иллюстрирующий продуцирование изопрена в Panteoa citrea, экспрессирующей pCL-lac-Kudzu. Серые ромбы представляют синтез изопрена, а черные квадраты представляют OD600.
На фигуре 10C представлен график, иллюстрирующий продуцирование изопрена в Panteoa citrea, экспрессирующей pTrcKudzu. Серые ромбы представляют синтез изопрена, а черные квадраты представляют OD600.
На фигуре 11 представлен график, иллюстрирующий продуцирование изопрена в клетках Bacillus subtilis, экспрессирующих рекомбинантную изопренсинтазу. BG3594comK представляет собой штамм B. subtilis, не содержащий плазмиды (продуцирующий нативный изопрен). CF443-BG3594comK представляет собой штамм B. subtilis, содержащий pBSKudzu (продуцирующий рекомбинантный изопрен). IS на оси Y означает изопрен.
На фигуре 12 представлена нуклеотидная последовательность pBS Kudzu #2 (SEQ ID NO:57).
На фигуре 13 представлена нуклеотидная последовательность изопренсинтазы пуэрарии, оптимизированная по кодонам для экспрессии в Yarrowia (SEQ ID NO: 8).
На фигуре 14 представлена карта плазмиды pTrex3g, содержащей ген изопренсинтазы пуэрарии, оптимизированный по кодонам для экспрессии в Yarrowia.
На фигуре 15 представлена нуклеотидная последовательность вектора pSPZ1(MAP29Spb) (SEQ ID NO:11).
На фигуре 16 представлена нуклеотидная последовательность синтетического гена изопрена пуэрарии (Pueraria montana), оптимизированного по кодонам для экспрессии в Yarrowia (SEQ ID NO: 12).
На фигуре 17 представлена нуклеотидная последовательность синтетического гена изопренсинтазы гибридного тополя (Populus alba x Populus tremula) (SEQ ID NO: 13). Старт-кодон ATG выделен жирным шрифтом, а стоп-кодон подчеркнут.
На фигуре 18A схематически представлен план конструирования векторов pYLA1, pYL1 и pYL2.
На фигуре 18B схематически представлен план конструирования вектора pYLA(POP1).
На фигуре 18С схематически представлен план конструирования вектора pYLA(KZ1).
На фигуре 18D схематически представлен план конструирования вектора pYLI(KZ1).
На фигуре 18E схематически представлен план конструирования вектора pYLI(MAP29).
На фигуре 18F схематически представлен план конструирования вектора pYLA(MAP29).
На фигуре 19 представлены метаболические пути MVA и DXP для изопрена (на основе публикации F. Bouvier et al., Progress in Lipid Res. 44: 357-429, 2005). Нижеследующее описание включает альтернативные названия для каждого полипептида в путях метаболизма, и работы, в которых описан анализ для измерения активности указанного полипептида (каждая из этих работ во всей своей полноте вводится в настоящее описание посредством ссылки, в частности, в описание анализов на полипептидную активность полипептидов, участвующих в путях MVA и DXP). Мевалонатный путь: AACT; ацетил-CoA-ацетилтрансфераза, MvaE, EC 2.3.1.9. Анализ: J. Bacteriol., 184: 2116-2122, 2002; HMGS; гидроксиметилглутарил-CoA-синтаза, MvaS, EC 2.3.3.10. Анализ: J. Bacteriol., 184: 4065-4070, 2002; HMGR; 3-гидрокси-3-метилглутарил-CoA-редуктаза, MvaE, EC 1.1.1.34. Анализ: J. Bacteriol., 184: 2116-2122, 2002; MVK; мевалонат-киназа, ERG12, EC 2.7.1.36. Анализ: Curr Genet 19:9-14, 1991. PMK; фосфомевалонат-киназа, ERG8, EC 2.7.4.2, Анализ: Mol. Cell Biol, 11:620-631, 1991; DPMDC; дифосфомевалонат-декарбоксилаза, MVD1, EC 4.1.1.33. Анализ: Biochemistry, 33:13355-13362, 1994; IDI; изопентенилдифосфат-дельта-изомераза, IDI1, EC 5.3.3.2. Анализ: J. Biol. Chem. 264:19169-19175, 1989. Путь DXР: DXS; 1-дезоксиксилулозо-5-фосфатсинтаза, dxs, EC 2.2.1.7. Анализ: PNAS, 94:12857-62, 1997; DXR; 1-дезокси-D-ксилулозо-5-фосфатредуктоизомераза, dxr, EC 2.2.1.7. Анализ: Eur. J. Biochem. 269:4446-4457, 2002; MCT; 4-дифосфоцитидил-2C-метил-D-эритритолсинтаза, IspD, EC 2.7.7.60. Анализ: PNAS, 97: 6451-6456, 2000; CMK; 4-дифосфоцитидил-2-C-метил-D-эритритолкиназа, IspE, EC 2.7.1.148. Анализ: PNAS, 97:1062-1067, 2000; MCS; 2C-метил-D-эритритол-2,4-циклодифосфатсинтаза, IspF, EC 4.6.1.12. Анализ: PNAS, 96:11758-11763, 1999; HDS; 1-гидрокси-2-метил-2-(E)-бутенил-4-дифосфатсинтаза, ispG, EC 1.17.4.3. Анализ: J. Org. Chem., 70:9168-9174, 2005; HDR; 1-гидрокси-2-метил-2-(E)-бутенил-4-дифосфатредуктаза, IspH, EC 1.17.1.2. Анализ: JACS, 126:12847-12855, 2004.
На фигуре 20 представлен график, иллюстрирующий результаты ЖХ-МС-анализа продуцирования изопрена рекомбинантными штаммами Y. lipolytica в отсутствии (слева) или в присутствии (справа) гена изопренсинтазы пуэрарии. Стрелками показано время элюирования аутентичного стандартного изопрена.
На фигуре 21 представлена карта pTrcKudzu yIDI DXS Kan.
На фигуре 22 представлена нуклеотидная последовательность pTrcKudzu yIDI DXS Kan (SEQ ID NO:20).
На фигуре 23A представлен график, иллюстрирующий продуцирование изопрена из глюкозы в BL21/pTrcKudzukan. Время 0 представляет собой время IPTG-индуцирования (400 мкмоль). На оси Х представлено время после индуцирования, на оси Y представлена OD600, а на оси Y2 представлена общая продуцируемость изопрена (мкг/л зоны «хэдспейс» или удельная продуцируемость (мкг/л зоны «хэдспейс»/OD)). Ромбы означают OD600, кружки означают общую продуцируемость изопрена (мкг/л), а квадраты означают удельное продуцирование изопрена (мкг/л/OD).
На фигуре 23B представлен график, иллюстрирующий продуцирование изопрена из глюкозы в BL21/pTrcKudzu yIDI kan. Время 0 представляет собой время IPTG-индуцирования (400 мкмоль). На оси Х представлено время после индуцирования, на оси Y представлена OD600, а на оси Y2 представлена общая продуцируемость изопрена (мкг/л зоны «хэдспейс» или удельная продуцируемость (мкг/л зоны «хэдспейс»/OD)). Ромбы означают OD600, кружки означают общую продуцируемость изопрена (мкг/л), а квадраты означают удельную продуцируемость изопрена (мкг/л/OD).
На фигуре 23C представлен график, иллюстрирующий продуцирование изопрена из глюкозы в BL21/pTrcKudzu DXS kan. Время 0 представляет собой время IPTG-индуцирования (400 мкмоль). На оси Х представлено время после индуцирования, на оси Y представлена OD600, а на оси Y2 представлена общая продуцируемость изопрена (мкг/л зоны «хэдспейс» или удельная продуцируемость (мкг/л зоны «хэдспейс»/OD)). Ромбы означают OD600, кружки означают общую продуцируемость изопрена (мкг/л), а квадраты означают удельную продуцируемость изопрена (мкг/л/OD).
На фигуре 23D представлен график, иллюстрирующий продуцирование изопрена из глюкозы в BL21/pTrcKudzu yIDI DXS kan. Время 0 представляет собой время IPTG-индуцирования (400 мкмоль). На оси Х представлено время после индуцирования, на оси Y представлена OD600, а на оси Y2 представлена общая продуцируемость изопрена (мкг/л зоны «хэдспейс» или удельная продуцируемость (мкг/л зоны «хэдспейс»/OD)). Ромбы означают OD600, кружки означают общую продуцируемость изопрена (мкг/л), а квадраты означают удельную продуцируемость изопрена (мкг/л/OD).
На фигуре 23E представлен график, иллюстрирующий продуцирование изопрена из глюкозы в BL21/pCL PtrcKudzu. Время 0 представляет собой время IPTG-индуцирования (400 мкмоль). На оси Х представлено время после индуцирования, на оси Y представлена OD600, а на оси Y2 представлена общая продуцируемость изопрена (мкг/л зоны «хэдспейс» или удельная продуцируемость (мкг/л зоны «хэдспейс»/OD)). Ромбы означают OD600, кружки означают общую продуцируемость изопрена (мкг/л), а квадраты означают удельную продуцируемость изопрена (мкг/л/OD).
На фигуре 23F представлен график, иллюстрирующий продуцирование изопрена из глюкозы в BL21/pCL PtrcKudzu yIDI. Время 0 представляет собой время IPTG-индуцирования (400 мкмоль). На оси Х представлено время после индуцирования, на оси Y представлена OD600, а на оси Y2 представлена общая продуцируемость изопрена (мкг/л зоны «хэдспейс» или удельная продуцируемость (мкг/л зоны «хэдспейс»/OD)). Ромбы означают OD600, кружки означают общую продуцируемость изопрена (мкг/л), а квадраты означают удельную продуцируемость изопрена (мкг/л/OD).
На фигуре 23G представлен график, иллюстрирующий продуцирование изопрена из глюкозы в BL21/pCL PtrcKudzu DXS. Время 0 представляет собой время IPTG-индуцирования (400 мкмоль). На оси Х представлено время после индуцирования, на оси Y представлена OD600, а на оси Y2 представлена общая продуцируемость изопрена (мкг/л зоны «хэдспейс» или удельная продуцируемость (мкг/л зоны «хэдспейс»/OD)). Ромбы означают OD600, кружки означают общую продуцируемость изопрена (мкг/л), а квадраты означают удельную продуцируемость изопрена (мкг/л/OD).
На фигуре 23H представлен график, иллюстрирующий продуцирование изопрена из глюкозы в BL21/pTrcKudzuIDIDXSkan. Стрелками показано время IPTG-индуцирования (400 мкмоль). На оси Х представлено время после индуцирования, на оси Y представлена OD600, а на оси Y2 представлена общая продуцируемость изопрена (мкг/л зоны «хэдспейс» или удельная продуцируемость (мкг/л зоны «хэдспейс»/OD)). Черные ромбы означают OD600, черные треугольники означают общую продуцируемость изопрена (мкг/л), а белые квадраты означают удельную продуцируемость изопрена (мкг/л/OD).
На фигуре 24 представлена карта pTrcKKDyIkIS kan.
На фигуре 25 представлена нуклеотидная последовательность pTrcKKDyIkIS kan (SEQ ID NO:33).
На фигуре 26 представлена карта pCL PtrcUpperPathway.
На фигурах 27A-27D представлена нуклеотидная последовательность pCL PtrcUpperPathway (SEQ ID NO:46).
На фигуре 28 представлена карта кластера, содержащего ген нижнего пути MVA и дрожжевой ген idi для интеграции в хромосому B. subtilis в локусе nprE. Вышерасположенный/нижерасположенный nprE означает 1 т.п.н. каждой последовательности локуса nprE для интеграции. Промотор гена aprE (промотор щелочной сериновой протеазы) означает промотор (сайт инициации транскрипции -35, -10, +1, RBS) гена aprE. MVK1 означает ген мевалонат-киназы дрожжей. RBS-PMK означает ген фосфомевалонат-киназы дрожжей, имеющий RBS Bacillus, расположенный выше старт-сайта. RBS-MPD означает ген дифосфомевалонат-декарбоксилазы дрожжей, имеющий RBS Bacillus, расположенный выше старт-сайта. RBS-IDI означает ген idi дрожжей, имеющий RBS Bacillus, расположенный выше старт-сайта. Терминатором является терминатор транскрипции гена щелочной сериновой протеазы от B. amyliquefaciens. SpecR означает маркер резистентности к спектиномицину. «Вышерасположенный nprE-повтор для ампл.» означает прямой повтор вышерасположенной области, используемой для амплификации.
На фигуре 29 представлена нуклеотидная последовательность кластера, содержащего ген нижнего пути MVA и дрожжевой ген idi для интеграции в хромосому B. subtilis в локусе nprE (SEQ ID NO:47).
На фигуре 30 представлена карта p9796 тополя.
На фигуре 31 представлена нуклеотидная последовательность p9796 тополя (SEQ ID NO:48).
На фигуре 32 представлена карта pTrcPOP1ar.
На фигуре 33 представлена нуклеотидная последовательность pTrcPOP1ar (SEQ ID NO:49).
На фигуре 34 представлена карта pTrcKudzu yIDI Kan.
На фигуре 35 представлена нуклеотидная последовательность pTrcKudzu yIDI Kan (SEQ ID NO: 50).
На фигуре 36 представлена нуклеотидная последовательность pTrcKudzuDXS Kan.
На фигуре 37 представлена нуклеотидная последовательность pTrcKudzuDXS Kan (SEQ ID NO: 51).
На фигуре 38 представлена карта pCL PtrcKudzu.
На фигуре 39 представлена нуклеотидная последовательность pCL PtrcKudzu (SEQ ID NO:52).
На фигуре 40 представлена карта pCL PtrcKudzu A3.
На фигуре 41 представлена нуклеотидная последовательность pCL PtrcKudzu A3 (SEQ ID NO:53).
На фигуре 42 представлена карта pCL PtrcKudzu yIDI.
На фигуре 43 представлена нуклеотидная последовательность pCL PtrcKudzu yIDI (SEQ ID NO:54).
На фигуре 44 представлена карта pCL PtrcKudzu DXS.
На фигуре 45 представлена нуклеотидная последовательность pCL PtrcKudzu DXS (SEQ ID NO:55).
На фигуре 46 представлены графики, иллюстрирующие продуцирование изопрена из кормовой биомассы. На панели A показано продуцирование изопрена из кукурузной соломы, на панели В показано продуцирование изопрена из багассы, на панели С показано продуцирование изопрена из пульпы мягкой древесины, на панели D показано продуцирование изопрена из глюкозы, а на панели Е показано продуцирование изопрена из клеток, не содержащих каких-либо дополнительных кормовых добавок. Серые квадраты означают величины OD600 культур в указанное время после инокуляции, а черные треугольники означают продуцирование изопрена в указанное время после инокуляции.
На фигуре 47A представлен график, иллюстрирующий продуцирование изопрена клетками BL21 (λDE3) pTrcKudzu yIDI DXS (kan) в культуре без добавления глюкозы. Квадраты означают OD600, а треугольники означают количество продуцированного изопрена (мкг/мл).
На фигуре 47B представлен график, иллюстрирующий продуцирование изопрена из 1% кормовой биомассы инвертированного сахара глюкозы клетками BL21 (λDE3) pTrcKudzu yIDI DXS (kan). Квадраты означают OD600, а треугольники означают количество продуцированного изопрена (мкг/мл).
На фигуре 47C представлен график, иллюстрирующий продуцирование изопрена из 1% кормовой биомассы инвертированного сахара клетками BL21 (λDE3) pTrcKudzu yIDI DXS (kan). Квадраты означают OD600, а треугольники означают количество продуцированного изопрена (мкг/мл).
На фигуре 47D представлен график, иллюстрирующий продуцирование изопрена из 1% кормовой биомассы кукурузной соломы AFEX клетками BL21 (λDE3) pTrcKudzu yIDI DXS (kan). Квадраты означают OD600, а треугольники означают количество продуцированного изопрена (мкг/мл).
На фигуре 48 представлены графики, иллюстрирующие влияние дрожжевого экстракта на продуцирование изопрена. На панели А указано количество времени, необходимое для достижения соответствующей оптической плотности в ферментере с подпиткой различными количествами дрожжевого экстракта. На панели В указано количество времени, необходимое для достижения соответствующего титра изопрена в ферментере с подпиткой различными количествами дрожжевого экстракта. Титр определяют как количество изопрена, продуцируемого на литр сбраживаемого бульона. На панели С проиллюстрировано влияние дрожжевого экстракта на уровень продуцирования изопрена в клетках E.coli, выращиваемых в периодической культуре с подпиткой.
На фигуре 49 представлены графики, иллюстрирующие продуцирование изопрена в 500-литровом биореакторе клетками E.coli, содержащими плазмиду pTrcKudzu+yIDI+DXS. На панели А указано количество времени, необходимое для достижения соответствующей оптической плотности в 500-литровом биореакторе с подпиткой глюкозой и дрожжевым экстрактом. На панели В указано количество времени, необходимое для достижения соответствующего титра изопрена в 500-литровом биореакторе с подпиткой глюкозой и дрожжевым экстрактом. Титр определяют как количество изопрена, продуцируемого на литр сбраживаемого бульона. На панели С указано количество времени, необходимое для продуцирования всего изопрена в 500-литровом биореакторе с подпиткой глюкозой и дрожжевым экстрактом.
На фигуре 50 представлена карта pJMupperpathway2.
На фигуре 51 представлена нуклеотидная последовательность pJMupperpathway2 (SEQ ID NO:56).
На фигуре 52 представлена карта pBS Kudzu #2.
На фигуре 53A представлен график, иллюстрирующий культивирование в процессе ферментации клеток Bacillus, экспрессирующих рекомбинантную изопренсинтазу пуэрарии в 14 литрах сбраживаемой периодической культуры с подпиткой. Черные ромбы означают контрольный штамм (BG3594comK), не содержащий рекомбинантной изопренсинтазы (продуцирующий нативный изопрен), а серые треугольники означают клетки Bacillus с pBSKudzu (продуцирующие рекомбинантный изопрен).
На фигуре 53B представлен график, иллюстрирующий продуцирование изопрена в процессе ферментации клеток Bacillus, экспрессирующих рекомбинантную изопренсинтазу пуэрарии в 14 литрах сбраживаемой периодической культуры с подпиткой. Черные ромбы означают контрольный штамм (BG3594comK), не содержащий рекомбинантной изопренсинтазы (продуцирующий нативный изопрен), а серые треугольники означают клетки Bacillus с pBSKudzu (продуцирующие рекомбинантный изопрен).
На фигуре 54 указано время, необходимое для достижения соответствующей оптической плотности в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 55 указано время, необходимое для достижения соответствующего титра изопрена в 15-литровом биореакторе с подпиткой глюкозой. Титр определяют как количество продуцируемого изопрена на литр сбраживаемого бульона.
На фигуре 56 указано время, необходимое для продуцирования всего изопрена в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 57 указано время, необходимое для достижения соответствующей оптической плотности в 15-литровом биореакторе с подпиткой глицерином.
На фигуре 58 указано время, необходимое для достижения соответствующего титра изопрена в 15-литровом биореакторе с подпиткой глицерином. Титр определяют как количество продуцируемого изопрена на литр сбраживаемого бульона.
На фигуре 59 указано время, необходимое для продуцирования всего изопрена в 15-литровом биореакторе с подпиткой глицерином.
На фигурах 60A-60C указано время, необходимое для достижения соответствующих оптической плотности, титра мевалоновой кислоты и удельной продуцируемости в 150-литровом биореакторе с подпиткой глюкозой.
На фигурах 61A-61C указано время, необходимое для достижения соответствующих оптической плотности, титра мевалоновой кислоты и удельной продуцируемости в 15-литровом биореакторе с подпиткой глюкозой.
На фигурах 62A-62C указано время, необходимое для достижения соответствующих оптической плотности, титра мевалоновой кислоты и удельной продуцируемости в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 63A-63C указано время, необходимое для достижения соответствующих оптической плотности, титра изопрена и удельной продуцируемости в 15-литровом биореакторе с подпиткой глюкозой.
На фигурах 64A-64C указано время, необходимое для достижения соответствующих оптической плотности, титра изопрена и удельной продуцируемости в 15-литровом биореакторе с подпиткой глюкозой.
На фигурах 65A-65C указано время, необходимое для достижения соответствующих оптической плотности, титра изопрена и удельной продуцируемости в 15-литровом биореакторе с подпиткой глюкозой.
На фигурах 66A-66C указано время, необходимое для достижения соответствующих оптической плотности, титра изопрена и удельной продуцируемости в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 67A-67C указано время, необходимое для достижения соответствующих оптической плотности, титра изопрена и удельной продуцируемости в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 68 представлен график вычисленных температур при адиабатическом воспламенении для серии А в зависимости от концентрации топлива для различных уровней кислорода. Надписи, приведенные на этой фигуре, соответствуют кривым в том порядке, в котором они представлены на графике. Так, например, первая надпись на этой фигуре (изопрен в воздухе при 40°С) соответствует самой высокой кривой на графике.
На фигуре 69 представлен график вычисленных температур при адиабатическом воспламенении для серии В в зависимости от концентрации топлива для различных уровней кислорода в присутствии 4% воды. Надписи, приведенные на этой фигуре, соответствуют кривым в том порядке, в котором они представлены на графике.
На фигуре 70 представлен график вычисленных температур при адиабатическом воспламенении для серии С в зависимости от концентрации топлива для различных уровней кислорода в присутствии 5% CO2. Надписи, приведенные на этой фигуре, соответствуют кривым в том порядке, в котором они представлены на графике.
На фигуре 71 представлен график вычисленных температур при адиабатическом воспламенении для серии D в зависимости от концентрации топлива для различных уровней кислорода в присутствии 10% CO2. Надписи, приведенные на этой фигуре, соответствуют кривым в том порядке, в котором они представлены на графике.
На фигуре 72 представлен график вычисленных температур при адиабатическом воспламенении для серии Е в зависимости от концентрации топлива для различных уровней кислорода в присутствии 15% CO2. Надписи, приведенные на этой фигуре, соответствуют кривым в том порядке, в котором они представлены на графике.
На фигуре 73 представлен график вычисленных температур при адиабатическом воспламенении для серии F в зависимости от концентрации топлива для различных уровней кислорода в присутствии 20% CO2. Надписи, приведенные на этой фигуре, соответствуют кривым в том порядке, в котором они представлены на графике.
На фигуре 74 представлен график вычисленных температур при адиабатическом воспламенении для серии G в зависимости от концентрации топлива для различных уровней кислорода в присутствии 30% CO2. Надписи, приведенные на этой фигуре, соответствуют кривым в том порядке, в котором они представлены на графике.
На фигуре 75A представлена таблица, иллюстрирующая превращение модели CAFT, результаты которого выражены как процент масса/объем для серии А.
На фигуре 75B представлен график результатов по воспламеняемости, полученных для модели CAFT серии А на фигуре 68 и выраженных как процент по объему.
На фигуре 76A представлена таблица, иллюстрирующая превращение модели CAFT, результаты которого выражены как процент масса/объем для серии В.
На фигуре 76B представлен график результатов по воспламеняемости, полученных для модели CAFT серии В на фигуре 69 и выраженных как процент по объему.
На фигуре 77 представлен сосуд для теста на воспламеняемость.
На фигуре 78A представлен график кривой воспламеняемости для серии испытаний 1: 0% пара, 0 фунт/кв. дюйм и 40°С.
На фигуре 78B представлена таблица, в которой систематизированы исходные данные по взрываемости и отсутствию взрываемости для серии испытаний 1.
На фигуре 78C представлен график кривой воспламеняемости для серии испытаний 1 по сравнению с моделью CAFT.
На фигуре 79A представлен график кривой воспламеняемости для серии испытаний 2: 0% пара, 0 фунт/кв. дюйм и 40°С.
На фигуре 79B представлена таблица, в которой систематизированы экспериментальные данные по взрываемости и отсутствию взрываемости для серии испытаний 2.
На фигуре 79C представлен график кривой воспламеняемости для серии испытаний 2 по сравнению с моделью CAFT.
На фигурах 80A и 80B представлены таблицы, в которых подробно описаны экспериментальные условия и результаты для серии испытаний 1.
На фигуре 81 представлены таблицы, в которых подробно описаны экспериментальные условия и результаты для серии испытаний 2.
На фигуре 82 представлен график вычисленных температур при адиабатическом воспламенении в зависимости от концентрации топлива для различных отношений азота/кислорода под давлением 3 атмосферы.
На фигуре 83 представлен график вычисленных температур при адиабатическом воспламенении в зависимости от концентрации топлива для различных отношений азота/кислорода под давлением в 1 атмосферу.
На фигуре 84 представлен график огибающей кривой воспламеняемости, который был построен с использованием данных, представленных на фигуре 82 и полученных методом, описанным в примере 13. Экспериментальные данные (кружки) были получены из тестов, описанных в настоящей заявке и проводимых под начальным давлением системы в 1 атмосферу.
На фигуре 85 представлен график огибающей кривой воспламеняемости, который был построен с использованием данных, представленных на фигуре 83 и полученных методом, описанным в примере 13. Экспериментальные данные (кружки) были получены из тестов, описанных в настоящей заявке и проводимых под начальным давлением системы в 1 атмосферу.
На фигуре 86A представлена ГХ/МС-хроматограмма отходящего газа при ферментации.
Фигура 86B является продолжением фигуры 86A, где, как это видно, присутствуют небольшие количества летучих веществ в отходящем газе при ферментации.
На фигуре 87A представлена ГХ/МС-хроматограмма следовых количеств летучих веществ в отходящем газе после крио-захвата при -78°С.
На фигуре 87B представлена ГХ/МС-хроматограмма следовых количеств летучих веществ в отходящем газе после крио-захвата при -196°C.
Фигура 87C является продолжением фигуры 87B.
Фигура 87D является продолжением фигуры 87C.
На фигурах 88A и 88B представлены ГХ/МС-хроматограммы, где приводится сравнение C5-углеводородов изопрена, полученного из нефти (фиг. 88A), и изопрена, полученного биологическим способом (фиг. 88B). Стандарт содержит три C5-углеводородных примеси, элюирующихся возле главного пика изопрена (фиг. 88A). В противоположность этому, изопрен, полученный биологическим способом, содержит некоторые количества этанола и ацетона (время реакции 3,41 минуты) (фиг. 88A).
На фигуре 89 представлен график, построенный по данным анализа отходящего газа при ферментации штамма BL21 (DE3) pTrcIS E.coli, экспрессирующего изопренсинтазу пуэрарии, где указанную ферментацию осуществляют с подпиткой глюкозой вместе с 3 г/л дрожжевого экстракта.
На фигуре 90 показаны структуры нескольких примесей, которые аналогичны изопрену и могут также действовать как вещества, отравляющие катализатор полимеризации.
На фигуре 91 представлена карта pTrcHis2AUpperPathway (также обозначаемая pTrcUpperMVA).
На фигурах 92A-92C представлена нуклеотидная последовательность плазмиды pTrcHis2AUpperPathway (также обозначаемой pTrcUpperMVA) (SEQ ID NO:86).
На фигуре 93 указано время, необходимое для достижения соответствующей оптической плотности в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 94 указано время, необходимое для достижения соответствующего титра изопрена в 15-литровом биореакторе с подпиткой глюкозой. Титр определяют как количество продуцируемого изопрена на литр сбраживаемого бульона.
На фигуре 95 указано время, необходимое для продуцирования всего изопрена в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 96 указано время, необходимое для достижения соответствующей оптической плотности в 15-литровом биореакторе с подпиткой инвертированным сахаром.
На фигуре 97 указано время, необходимое для достижения соответствующего титра изопрена в 15-литровом биореакторе с подпиткой инвертированным сахаром. Титр определяют как количество продуцируемого изопрена на литр сбраживаемого бульона.
На фигуре 98 указано время, необходимое для продуцирования всего изопрена в 15-литровом биореакторе с подпиткой инвертированным сахаром.
На фигуре 99 указано время, необходимое для достижения соответствующей оптической плотности в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 100 указано время, необходимое для достижения соответствующего титра изопрена в 15-литровом биореакторе с подпиткой глюкозой. Титр определяют как количество продуцируемого изопрена на литр сбраживаемого бульона.
На фигуре 101 указано время, необходимое для достижения удельной активности изопрена в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 102 представлена карта pCLPtrcUpperPathwayHGS2.
На фигурах 103A-103C представлена нуклеотидная последовательность pCLPtrcUpperPathwayHGS2 (SEQ ID NO: 87).
На фигуре 104 указано время, необходимое для достижения соответствующей оптической плотности в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 105 указано время, необходимое для достижения соответствующего титра изопрена в 15-литровом биореакторе с подпиткой глюкозой. Титр определяют как количество продуцируемого изопрена на литр сбраживаемого бульона.
На фигуре 106 указано время, необходимое для продуцирования всего изопрена в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 107 представлена карта плазмиды MCM330.
На фигурах 108A-108C представлена нуклеотидная последовательность плазмиды MCM330 (SEQ ID NO:90).
На фигуре 109 представлена карта pET24D-Kudzu.
На фигурах 110A и 110B представлена нуклеотидная последовательность pET24D-Kudzu (SEQ ID NO: 101).
На фигуре 111A указано время, необходимое для достижения соответствующей оптической плотности в 15-литровом биореакторе с подпиткой глюкозой.
На фигуре 111В указано время, необходимое для достижения соответствующего титра изопрена в 15-литровом биореакторе с подпиткой глюкозой. Титр определяют как количество продуцируемого изопрена на литр сбраживаемого бульона.
На фигуре 111С указано время, необходимое для достижения удельной продуцируемости изопрена в 15-литровом биореакторе с подпиткой глюкозой.
Подробное описание изобретения
В одном из своих аспектов, настоящее изобретение относится к композициям для продуцирования и к способам продуцирования изопрена в более высоких количествах и/или с более высокой степенью чистоты. Используемый здесь термин «изопрен» или «2-метил-1,3-бутадиен» (CAS# 78-79-5) означает прямой и конечный летучий C5-углеводородный продукт, образующийся в результате элиминации пирофосфата из 3,3-диметилаллилпирофосфата (DMAPP), но не в результате связывания или полимеризации одной или более молекул изопентенилдифосфата (IPP) с одной или более молекулами DMAPP.
В большинстве случаев, изопрен получают из нефтехимических источников в виде примесной C5-углеводородной фракции, и для получения материала, подходящего для полимеризации, эта фракция требует тщательной очистки. Такая очистка представляет серьезные трудности, так как некоторые примеси имеют структурное сходство с изопреном, и они могут действовать как вещества, отравляющие катализатор полимеризации. Такими соединениями являются 1,3-циклопентадиен, цис- и транс-1,3-пентадиен, 1-пентин, 2-пентин, 3-метил-1-бутин, транс-пиперилен, цис-пиперилен, пент-4-ен-1-ин, транс-пент-3-ен-1-ин или цис-пент-3-ен-1-ин (фиг. 90). В некоторых вариантах изобретения, изопреновая композиция согласно изобретению, в основном, не содержит каких-либо примесных ненасыщенных C5-углеводородов. Как более подробно описано в примере 10, в композициях изопрена, полученных описанными здесь способами, не было обнаружено какого-либо детектируемого количества ненасыщенных C5-углеводородов, не являющихся изопреном (таких как 1,3-циклопентадиен, цис-1,3-пентадиен, транс-1,3-пентадиен, 1-пентин, 2-пентин, 1-пентен, 2-метил-1-бутен, 3-метил-1-бутин, транс-пиперилен, цис-пиперилен, пент-4-ен-1-ин, транс-пент-3-ен-1-ин или цис-пент-3-ен-1-ин). Некоторые композиции изопрена, полученные описанными здесь методами, содержат этанол, ацетон и прениловые С5-спирты, как было определено с помощью ГХ/МС-анализа. Все эти компоненты гораздо легче удалить из изопренового пара, чем из изомерных С5-углеводородных фракций, присутствующих в изопреновых композициях, полученных их нефтехимических источников. В соответствии с этим, в некоторых вариантах изобретения, для достижения нужной степени полимеризации требуется лишь минимальная обработка изопреновых композиций согласно изобретению.
В одном из аспектов изобретения, композиции и способы согласно изобретению позволяют увеличивать скорость продуцирования изопрена и увеличивать общее количество продуцируемого изопрена. Так, например, были продуцированы системы культивирования клеток, генерирующие 4,8×104 нмоль/гwcm/час изопрена (таблица 1). Эффективность этих систем была продемонстрирована на примере превращения приблизительно 2,2% углерода, который потребляют клетки из культуральной клеточной среды, в изопрен. Как показано в примерах и в таблице 2, было получено приблизительно 3 г изопрена на литр бульона. При желании, может быть получено еще большее количество изопрена в других условиях, таких как условия, описанные в настоящей заявке. В некоторых вариантах изобретения, для продуцирования изопрена используют возобновляемый источник углерода. В некоторых вариантах изобретения, продуцирование изопрена не связано с ростом клеток. В некоторых вариантах изобретения, для снижения или устранения риска воспламенения, которое может происходить в процессе продуцирования или регенерации изопрена, изопрен и любые окислители должны присутствовать в концентрациях, не выходящих за пределы невоспламеняемости. Композиции и способы согласно изобретению являются предпочтительными, поскольку они дают высокий выход изопрена на клетку, высокий выход углерода, высокую чистоту изопрена, имеют высокую продуктивность и низкое потребление энергии; не требуют больших затрат и инвестиций, и дают минимальные побочные эффекты. Такой эффективный промышленный способ биосинтеза изопрена позволяет получить источник изопрена для синтеза изопренового каучука и представляет собой желательную экономически выгодную альтернативу использованию природного каучука.
Как обсуждается ниже, количество изопрена, продуцируемого клетками, может быть значительно увеличено путем введения в клетки гетерологичной нуклеиновой кислоты, кодирующей полипептид изопренсинтазы (например, полипептид изопренсинтазы растений). Полипептиды изопренсинтазы превращают диметилаллилдифосфат (DMAPP) в изопрен. Как показано в примерах, гетерологичный полипептид изопренсинтазы Pueraria Montana (пуэрарии волосистой) экспрессируется в различных клетках-хозяевах, таких как Escherichia coli, Panteoa citrea, Bacillus subtilis, Yarrowia lipolytica и Trichoderma reesei. Все эти клетки продуцируют большее количество изопрена, чем соответствующие клетки, не содержащие гетерологичного полипептида изопренсинтазы. Как проиллюстрировано в таблицах 1 и 2, большие количества изопрена получают описанными здесь способами. Так, например, клетки B. subtilis, содержащие гетерологичную нуклеиновую кислоту изопренсинтазы, продуцируют приблизительно в 10 раз большее количество изопрена в 14-литровом ферментере, чем соответствующие контрольные клетки B. subtilis, не содержащие такой гетерологичной нуклеиновой кислоты (таблица 2). Продуцирование 300 мг изопрена на литр бульона (мг/л, где объем бульона включает объем клеточной среды и объем клеток) клетками E.coli и 30 мг/л клетками B. subtilis в ферментерах указывает на то, что может быть получено значительное количество изопрена (таблица 2). При желании, изопрен может быть получен в еще большем масштабе, либо для еще большего увеличения количества изопрена могут быть использованы другие описанные здесь условия. Векторы, перечисленные в таблицах 1 и 2, и экспериментальные условия более подробно описаны ниже и в разделе «Примеры».
Таблица 1: Репрезентативные выходы изопрена из шейкерной колбы, полученные с использованием клеточных культур и с применением способов согласно изобретению
Анализ, проводимый для измерения уровня продуцирования изопрена, описан в примере I, часть II. Для этого анализа, образец брали из шейкерной колбы через определенные один или более интервалов времени, и культивировали в течение 30 минут. Затем измеряли количество изопрена, продуцируемого в этом образце. Концентрации изопрена в зоне «хэдспейс» и константы скорости продуцирования изопрена указаны в таблице 1 и более подробно описаны ниже.
(нмоль/гwcm/час)
Таблица 2: Репрезентативные выходы изопрена в ферментере, полученные с использованием клеточных культур и с применением способов согласно изобретению
Анализ, проводимый для измерения уровня продуцирования изопрена, описан в примере I, часть II. Для этого анализа брали образец отходящего газа из ферментера, и этот образец анализировали на количество изопрена. Максимальная концентрация в зоне «хэдспейс» (которая представляет собой наивысшую концентрацию в зоне «хэдспейс» в процессе ферментации), титр (который представляет собой кумулятивное общее количество продуцируемого изопрена на литр бульона) и максимальная константа скорости продуцирования изопрена (которая представляет собой наивысшую константу скорости в процессе ферментации) указаны в таблице 2 и более подробно описаны ниже.
(нмоль/гwcm/час)
2-pTrcKKDyIkIS
Кроме того, продуцирование изопрена клетками, содержащими гетерологичную нуклеиновую кислоту изопренсинтазы, может быть повышено путем увеличения количества полипептида 1-дезокси-D-ксилулозо-5-фосфатсинтазы (DXS) и/или полипептида изопентенилдифосфатизомеразы (IDI), экспрессируемых клетками. Так, например, нуклеиновая кислота DXS и/или нуклеиновая кислота IDI могут быть введены в клетки. Нуклеиновой кислотой DXS может быть гетерологичная нуклеиновая кислота или дуплицированная копия эндогенной нуклеиновой кислоты. Аналогичным образом, нуклеиновой кислотой IDI может быть гетерологичная нуклеиновая кислота или дуплицированная копия эндогенной нуклеиновой кислоты. В некоторых вариантах изобретения, количество полипептида DXS и/или IDI повышают путем замены эндогенных промоторов или регуляторных областей DXS и/или IDI другими промоторами и/или другими регуляторными областями, повышающими уровень транскрипции нуклеиновых кислот DXS и/или IDI. В некоторых вариантах изобретения, клетки содержат гетерологичную нуклеиновую кислоту, кодирующую полипептид изопренсинтазы (например, нуклеиновую кислоту изопренсинтазы растений), и дуплицированную копию эндогенной нуклеиновой кислоты, кодирующей полипептид изопренсинтазы.
Кодируемые полипептиды DXS и IDI являются частью пути DXP биосинтеза изопрена (фигура 19). Полипептиды DXS превращают пируват и D-глицеральдегид-3-фосфат в 1-дезокси-D-ксилулозо-5-фосфат. Не ограничиваясь какой-либо конкретной теорией, следует отметить, что с увеличением количества полипептида DXS увеличивается поток углерода по пути DXP, что приводит к усилению продуцирования изопрена. Полипептиды IDI катализируют взаимное превращение изопентенилдифосфата (IPP) и диметилаллилдифосфата (DMAPP). Не ограничиваясь какой-либо конкретной теорией, следует отметить, что с увеличением количества полипептида IDI в клетках увеличивается количество (и повышается скорость превращения) IPP, который превращается в DMAPP, который, в свою очередь превращается в изопрен.
Так, например, для продуцирования изопрена, клетки E.coli подвергают ферментации с использованием нуклеиновых кислот изопренсинтазы пуэрарии, IDI S. cerevisia и DXS E.coli. Уровни изопрена варьировались от 50 до 300 мкг/л в течение периода времени 15 часов (пример 7, часть VII).
В некоторых вариантах изобретения, присутствие гетерологичных или дополнительных эндогенных нуклеиновых кислот изопренсинтазы, IDI и DXS стимулирует более репродуцируемый рост клеток или позволяет им оставаться жизнеспособными в течение более длительного периода времени по сравнению с соответствующими клетками, содержащими только одну или две из этих гетерологичных или дополнительных эндогенных нуклеиновых кислот. Так, например, клетки, содержащие гетерологичные нуклеиновые кислоты изопренсинтазы, IDI и DXS, растут лучше, чем клетки, имеющие только гетерологичные нуклеиновые кислоты изопренсинтазы и DXS, или только гетерологичную нуклеиновую кислоту изопренсинтазы. Кроме того, гетерологичные нуклеиновые кислоты изопренсинтазы, IDI и DXS были успешно функционально присоединены к сильному промотору на высококопийной плазмиде, которая сохранялась в клетках E.coli, что дает основание предположить, что большие количества этих полипептидов могут экспрессироваться в клетках, не оказывая какого-либо избыточного токсического воздействия на эти клетки. Не ограничиваясь какой-либо конкретной теорией, следует отметить, что присутствие гетерологичной или дополнительной эндогенной нуклеиновой кислоты изопренсинтазы и IDI может приводить к снижению количества одного или более потенциально токсичных промежуточных соединений, которые так или иначе могут накапливаться в случае, если в клетках будет присутствовать только гетерологичная или дополнительная эндогенная нуклеиновая кислота DXS.
В некоторых вариантах изобретения, продуцирование изопрена клетками, содержащими гетерологичную нуклеиновую кислоту изопренсинтазы, увеличивается с увеличением количества полипептида MVA, экспрессируемого клетками (фигура 19). Репрезентативными полипептидами путей MVA являются любые нижеследующие полипептиды: полипептиды ацетил-CoA ацетилтрансферазы (AA-CoA-тиолазы), полипептиды 3-гидрокси-3-метилглутарил-CoA-синтазы (HMG-CoA-синтазы), полипептиды 3-гидрокси-3-метилглутарил-CoA-редуктазы (HMG-CoA-редуктазы), полипептиды мевалонаткиназы (MVK), полипептиды фосфомевалонаткиназы (PMK), полипептиды дифосфомевалонатдекарбоксилазы (MVD), полипептиды IDI и полипептиды (например, гибридные полипептиды), обладающие активностью двух или более полипептидов пути MVA. Так, например, в клетки могут быть введены одна или более нуклеиновых кислот, кодирующих полипептиды пути MVA. В некоторых вариантах изобретения, клетки содержат нуклеиновые кислоты, кодирующие полипептиды верхнего пути MVA, которые включают нуклеиновые кислоты, кодирующие AA-CoA-тиолазу, HMG-CoA-синтазу и HMG-CoA-редуктазу. В некоторых вариантах изобретения, клетки содержат нуклеиновые кислоты, кодирующие полипептиды нижнего пути MVA, которые включают нуклеиновые кислоты MVK, PMK, MVD и IDI. В некоторых вариантах изобретения, клетки содержат нуклеиновые кислоты, кодирующие полипептиды полного пути MVA, где указанными нуклеиновыми кислотами являются нуклеиновые кислоты AA-CoA-тиолазы, HMG-CoA-синтазы, HMG-CoA-редуктазы, MVK, PMK, MVD и IDI. Нуклеиновыми кислотами пути MVA могут быть гетерологичные нуклеиновые кислоты или дуплицированные копии эндогенных нуклеиновых кислот. В некоторых вариантах изобретения, количество одного или более полипептидов пути MVA повышают путем замены эндогенных промоторов или регуляторных областей нуклеиновых кислот пути MVA другими промоторами и/или регуляторными областями, которые повышают уровень транскрипции нуклеиновых кислот пути MVA. В некоторых вариантах изобретения, клетки содержат гетерологичную нуклеиновую кислоту, кодирующую полипептид изопренсинтазы (например, нуклеиновую кислоту растительной изопренсинтазы), и дуплицированную копию эндогенной нуклеиновой кислоты, кодирующей полипептид изопренсинтазы.
Так, например, клетки E.coli, содержащие нуклеиновую кислоту, кодирующую полипептид изопренсинтазы пуэрарии, и нуклеиновую кислоту, кодирующую полипептиды MVK, PMK, MVD и IDI Saccharomyces cerevisia, продуцировали изопрен со скоростью 6,67×10-4 моль/лбульона/OD600/час (см. пример 8). Кроме того, при ферментации в 14-литровом масштабе, клетки E.coli, имеющие нуклеиновую кислоту, кодирующую полипептиды AA-CoA-тиолазы, HMG-CoA-синтазы и HMG-CoA-редуктазы Enterococcus faecalis, продуцировали 22 грамма мевалоновой кислоты (промежуточное соединение пути MVA). Клетки в шейкерных колбах продуцировали 2-4 грамма мевалоновой кислоты на литр. Эти результаты показали, что гетерологичные нуклеиновые кислоты путей MVA являются активными в E.coli. Клетки E.coli, содержащие нуклеиновые кислоты верхнего пути MVA и нижнего пути MVA, а также изопренсинтазы пуэрарии (штамм MCM 127), продуцировали значительно большее количество изопрена (874 мкг/л) по сравнению с клетками E.coli, содержащими нуклеиновые кислоты только нижнего пути MVA и изопренсинтазы пуэрарии (штамм MCM 131) (см. таблицу 3 и пример 8, часть VIII).
В некоторых вариантах изобретения, по меньшей мере часть клеток сохраняет гетерологичную нуклеиновую кислоту изопренсинтазы, DXS, IDI, и/или пути MVA по меньшей мере приблизительно за 5, 10, 20, 50, 75, 100, 200, 300 или более делений клеток в непрерывной культуре (такой как непрерывная культура без разведения). В некоторых вариантах любого из аспектов изобретения, нуклеиновая кислота, содержащая гетерологичную или дуплицированную копию эндогенной нуклеиновой кислоты изопренсинтазы, DXS, IDI и/или пути MVA, также содержит селективный маркер, такой как маркер резистентности нуклеиновой кислоты к антибиотикам, таким как канамицин, ампициллин, карбенициллин, гентамицин, гигромицин, флеомицин, блеомицин, неомицин или хлорамфеникол.
Как указано в примере 7, части VI, количество продуцируемого изопрена может быть также увеличено путем добавления дрожжевого экстракта в среду для культивирования клеток. В этом примере, количество продуцируемого изопрена прямо пропорционально количеству дрожжевого экстракта в клеточной среде в тестируемых концентрациях (фигура 48C). Кроме того, приблизительно 0,11 грамма изопрена на литр бульона было продуцировано из клеточной среды, содержащей дрожжевой экстракт и глюкозу (пример 7, часть VIII). В обоих этих экспериментах, для продуцирования изопрена использовали клетки E.coli, содержащие нуклеиновые кислоты изопренсинтазы пуэрарии, IDI S. cerevisia и DXS E.coli. Увеличение количества дрожжевого экстракта в присутствии глюкозы приводит к увеличению уровня продуцирования изопрена по сравнению с уровнем, наблюдаемым при увеличении количества глюкозы в присутствии дрожжевого экстракта. Кроме того, увеличение количества дрожжевого экстракта позволяет клеткам продуцировать высокий уровень изопрена в течение более длительного периода времени и повышает жизнеспособность клеток.
Продуцирование изопрена также продемонстрировано на примере гидролизованной биомассы трех типов (багассы, кукурузной соломы и пульпы мягкой древесины), используемой в качестве источника углерода (фигуры 46 A-C). Клетки E.coli, содержащие нуклеиновые кислоты изопренсинтазы пуэрарии, IDI S. cerevisia и DXS E.coli, продуцировали такое же большое количество изопрена из этой гидролизованной биомассы, используемой в качестве источника углерода, как и количество, продуцируемое из эквивалентного количества глюкозы (например, 1% глюкозы, масс/об). При желании, в композициях и в способах согласно изобретению, в качестве источника углерода может быть использована и другая биомасса. Биомасса как источник углерода является более предпочтительной, поскольку она дешевле многих стандартных клеточных сред, что позволяет осуществлять более экономичное продуцированиe изопрена.
Кроме того, было показано, что инвертированный сахар служит источником углерода при продуцировании изопрена (фигуры 47C и 96-98). Так, например, из клеток, экспрессирующих полипептиды пути MVA и изопренсинтазу пуэрарии, продуцируется 2,4 г/л изопрена (пример 8, часть XV). Для продуцирования 2,2 мг/л изопрена из клеток, экспрессирующих изопренсинтазу пуэрарии, в качестве источника углерода также использовался глицерин (пример 8, часть XIV). Экспрессия нуклеиновой кислоты DXS, нуклеиновой кислоты IDI и/или одной или более нуклеиновых кислот пути MVA (таких как нуклеиновые кислоты, кодирующие весь путь MVA), помимо экспрессии нуклеиновой кислоты изопренсинтазы, может приводить к повышению уровня продуцирования изопрена из глицерина.
В некоторых вариантах изобретения, в клеточную среду включают масло. Так, например, клетки B. subtilis, содержащие нуклеиновую кислоту изопренсинтазы пуэрарии, продуцируют изопрен при их культивировании в клеточной среде, содержащей масло и источник глюкозы (пример 4, часть III). В некоторых вариантах изобретения, в клеточную среду включают более чем одно масло (например, 2, 3, 4, 5 или более масел). Не ограничиваясь какой-либо конкретной теорией, следует отметить, что (i) масло может увеличивать количество углерода в клетках, который является доступным для превращения в изопрен, (ii) масло может увеличивать количество ацетил-CoA в клетках, и тем самым, увеличивать поток углерода по пути MVA, и/или (ii) масло может снабжать клетки дополнительными микроэлементами, что является желательным, поскольку большая часть углерода в клетках превращается в изопрен, а не в другие продукты. В некоторых вариантах изобретения, клетки, которые культивируют в клеточной среде, содержащей масло, для продуцирования изопрена обычно используют путь MVA, либо эти клетки генетически модифицируют так, чтобы они содержали нуклеиновые кислоты всего пути MVA. В некоторых вариантах изобретения, такое масло частично или полностью гидролизуют, а затем добавляют в культуральную клеточную среду для облегчения утилизации масла клетками-хозяевами.
Одной из главных проблем при промышленном продуцировании небольших молекул, таких как изопрен в клетках (например, в бактериях), является нарушение взаимосвязи между продуцированием такой молекулы и ростом клеток. В некоторых вариантах коммерчески эффективного продуцирования изопрена, значительное количество углерода в кормовой биомассе превращается в изопрен, но не стимулирует рост и поддержание жизнеспособности клеток («выход углерода»). В различных вариантах изобретения, клетки превращают более чем или примерно 0,0015, 0,002, 0,005, 0,01, 0,02, 0,05, 0,1, 0,12, 0,14, 0,16, 0,2, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9, 1,0, 1,2, 1,4, 1,6, 1,8, 2,0, 2,5, 3,0, 3,5, 4,0, 5,0, 6,0, 7,0 или 8,0% углерода в культуральной клеточной среде в изопрен. В конкретных вариантах изобретения, значительная часть углерода кормовой биомассы, превращаемой в последующие продукты, превращается в изопрен. Как подробно описано в примере 11, по мере роста клеток E.coli, экспрессирующих нуклеиновые кислоты пути MVA и нуклеиновые кислоты изопренсинтазы пуэрарии, продуцирование в них изопрена или промежуточной мевалоновой кислоты ослабляется, что приводит к продуцированию высокого выхода углерода. В частности, в клетках, экспрессирующих верхний путь MVA Enterococcus faecalis, образуется мевалоновая кислота. Изопрен образуется в клетках, экспрессирующих верхний путь MVA Enterococcus faecalis, нижний путь MVA Saccharomyces cerevisiae и изопренсинтазу Pueraria montana (пуэрарии волосистой). Такое нарушение прямой взаимосвязи между продуцированием изопрена или мевалоновой кислоты и ростом клеток наблюдалось в четырех различных штаммах клеток E.coli: BL21(LDE3), клеток-переключателей BL21(LDE3), клеток FM5 и клеток MG1655. Первые два штамма E.coli представляют собой штаммы B, а другие два штамма представляют собой штаммы K12. Ослабление продуцирования указанных соединений по мере роста клеток также было продемонстрировано для варианта MG 1655, в котором были делетированы гены ack и pta. Этот вариант также обнаруживал меньший уровень продуцирования ацетата.
Репрезентативные полипептиды и нуклеиновые кислоты
В композициях и способах согласно изобретению могут быть использованы различные полипептиды изопренсинтазы, DXS, IDI и/или пути MVA и нуклеиновые кислоты, кодирующие эти полипептиды.
Используемый здесь термин «полипептиды» включает полипептиды, белки, пептиды, фрагменты полипептидов и гибридные полипептиды. В некоторых вариантах изобретения, указанные гибридные полипептиды включают часть первого полипептида или весь первый полипептид (например, полипептид изопренсинтазы, DXS, IDI или полипептид пути MVA) и часть второго полипептида или весь второй полипептид (например, пептид, который облегчает очистку или детектирование гибридного полипептида, такой как His-метка). В некоторых вариантах изобретения, гибридный полипептид обладает активностью двух или более полипептидов пути MVA (таких как полипептиды AA-CoA-тиолазы и HMG-CoA-редуктазы). В некоторых вариантах изобретения, указанным полипептидом является природный полипептид (такой как полипептид, кодируемый нуклеиновой кислотой mvaE Enterococcus faecalis), который обладает активностью двух или более полипептидов пути MVA.
В различных вариантах изобретения, полипептид имеет по меньшей мере или примерно 50, 100, 150, 175, 200, 250, 300, 350, 400 или более аминокислот. В некоторых вариантах изобретения, полипептидный фрагмент содержит по меньшей мере или примерно 25, 50, 75, 100, 150, 200, 300 или более смежных аминокислот, происходящих от полноразмерного полипептида, и обладает активностью, которая составляет по меньшей мере или примерно 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95% или 100% от активности соответствующего полноразмерного полипептида. В конкретных вариантах изобретения, указанный полипептид включает сегмент аминокислотной последовательности или всю аминокислотную последовательность любого природного полипептида изопренсинтазы, DXS, IDI или пути MVA. В некоторых вариантах изобретения, указанный полипептид, по сравнению с последовательностью полипептида изопренсинтазы, DXS, IDI или пути MVA дикого типа (то есть с природной последовательностью), имеет одну или более мутаций.
В некоторых вариантах изобретения, указанным полипептидом является выделенный полипептид. Используемый здесь термин «выделенный полипептид» не является частью библиотеки полипептидов, такой как библиотека из 2, 5, 10, 20, 50 или более различных полипептидов, и отделен по меньшей мере от одного компонента, с которым он встречается в природе. Выделенный полипептид может быть получен, например, путем экспрессии рекомбинантной нуклеиновой кислоты, кодирующей данный полипептид.
В некоторых вариантах изобретения, указанным полипептидом является гетерологичный полипептид. Термин «гетерологичный полипептид» означает полипептид, аминокислотная последовательность которого не идентична аминокислотной последовательности другого полипептида, обычно экспрессируемого той же самой клеткой-хозяином. В частности, гетерологичный полипептид не идентичен полипептиду, кодируемому нуклеиновой кислотой дикого типа, присутствующей в природе в той же самой клетке-хозяине.
Используемый здесь термин «нуклеиновая кислота» означает два или более дезоксирибонуклеотидов и/или рибонуклеотидов, присутствующих в одноцепочечной или двухцепочечной форме. В некоторых вариантах изобретения, нуклеиновой кислотой является рекомбинантная нуклеиновая кислота. Термин «рекомбинантная нуклеиновая кислота» означает представляющую интерес нуклеиновую кислоту, которая не содержит одной или более нуклеиновых кислот (например, генов), присутствующих в геноме природного организма, от которого происходит данная представляющая интерес нуклеиновая кислота, и фланкирующих данную представляющую интерес нуклеиновую кислоту. Поэтому этот термин включает, например, рекомбинантную ДНК, которая встроена в вектор, в автономно реплицирующуюся плазмиду или вирус, или в геномную ДНК прокариота или эукариота, или которая существует как отдельная молекула (например, кДНК, фрагмент геномной ДНК или фрагмент кДНК, продуцируемые с помощью ПЦР или путем гидролиза рестриктирующей эндонуклеазой), не связанная с другими последовательностями. В различных вариантах изобретения, указанной нуклеиновой кислотой является рекомбинантная нуклеиновая кислота. В некоторых вариантах изобретения, нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA функционально присоединена к другой нуклеиновой кислоте, кодирующей полноразмерный другой полипептид или его часть так, что в результате этого указанная рекомбинантная нуклеиновая кислота кодирует гибридный полипептид, который включает полипептид изопренсинтазы, DXS, IDI или пути MVA и полноразмерный другой полипептид или его часть (например, пептид, облегчающий очистку или детектирование гибридного полипептида, такой как His-метка). В некоторых вариантах изобретения, часть рекомбинантной нуклеиновой кислоты или вся рекомбинантная нуклеиновая кислота являются химически синтезированными.
В некоторых вариантах изобретения, указанной нуклеиновой кислотой является гетерологичная нуклеиновая кислота. Термин «гетерологичная нуклеиновая кислота» означает нуклеиновую кислоту, последовательность которой не идентична последовательности другой нуклеиновой кислоты, обычно присутствующей в той же самой клетке-хозяине.
В конкретных вариантах изобретения, нуклеиновая кислота включает сегмент последовательности или полноразмерную последовательность любой природной нуклеиновой кислоты изопренсинтазы, DXS, IDI или пути MVA. В некоторых вариантах изобретения, нуклеиновая кислота включает по меньшей мере или примерно 50, 100, 150, 200, 300, 400, 500, 600, 700, 800 или более смежных нуклеотидов, происходящих от природной нуклеиновой кислоты изопренсинтазы, DXS, IDI или пути MVA. В некоторых вариантах изобретения, нуклеиновая кислота, в отличие от последовательности нуклеиновой кислоты изопренсинтазы, DXS, IDI или пути MVA дикого типа (то есть природной последовательности), имеет одну или более мутаций. В некоторых вариантах изобретения, нуклеиновая кислота имеет одну или более мутаций (например, молчащих мутаций), которые способствуют повышению уровня транскрипции или трансляции нуклеиновой кислоты изопренсинтазы, DXS, IDI или пути MVA. В некоторых вариантах изобретения, указанной нуклеиновой кислотой является вырожденный вариант любой нуклеиновой кислоты, кодирующей полипептид изопренсинтазы, DXS, IDI или пути MVA.
Термин «вырожденность кодона» означает расхождение в генетическом коде, приводящее к изменению нуклеотидной последовательности, но не влияющее на аминокислотную последовательность кодируемого полипептида. Специалисту в данной области хорошо известно о «смещении кодонов», наблюдаемом в конкретных клетках-хозяевах, в которых используются нуклеотидные кодоны, специфичные для данной аминокислоты. Поэтому, в некоторых вариантах изобретения, при синтезе нуклеиновой кислоты, осуществляемом в целях улучшения экспрессии в клетке-хозяине, желательно сконструировать такую нуклеиновую кислоту, в которой частота встречаемости кодонов будет приближаться к частоте встречаемости предпочтительных кодонов в клетке-хозяине.
Регистрационные номера репрезентативных полипептидов изопренсинтазы, DXS, IDI, и/или пути MVA и кодирующих их нуклеиновых кислот перечислены в Приложении 1 настоящей заявки, и их соответствующие последовательности во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в разделе, относящемся к аминокислотным последовательностям и нуклеотидным последовательностям полипептидов изопренсинтазы, DXS, IDI, и/или пути MVA. База данных Kegg также содержит аминокислотные и нуклеотидные последовательности различных репрезентативных полипептидов изопренсинтазы, DXS, IDI, и/или MVA (см., например, сайт в Интернете «genome.jp/kegg/pathway/map/map00100.html» и имеющиеся там последовательности, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание аминокислотных последовательностей и нуклеотидных последовательностей полипептидов и нуклеиновых кислот изопренсинтазы, DXS, IDI, и/или пути MVA). В некоторых вариантах изобретения, один или более полипептидов изопренсинтазы, DXS, IDI, и/или пути MVA и/или нуклеиновых кислот имеют последовательности, идентичные последовательностям, имеющимся в базе данных, которая стала общедоступной 12 декабря 2007 г., например, любые последовательности, соответствующие последовательностям, имеющим любой из регистрационных номеров в Приложении 1 настоящей заявки, или любые последовательности, представленные в базе данных Kegg на день подачи данной заявки. Другие репрезентативные полипептиды и нуклеиновые кислоты изопренсинтазы, DXS, IDI, и/или пути MVA более подробно описаны ниже.
Репрезентативные полипептиды и нуклеиновые кислоты изопренсинтазы
Как указывалось выше, полипептиды изопренсинтазы превращают диметилаллилдифосфат (DMAPP) в изопрен. Репрезентативными полипептидами изопренсинтазы являются полипептиды, фрагменты полипептидов, пептиды и гибридные полипептиды, обладающие по меньшей мере одной активностью полипептида изопренсинтазы. Для того, чтобы определить, обладает ли полипептид активностью полипептида изопренсинтазы, могут быть применены стандартные методы, проводимые путем оценки способности полипептида превращать DMAPP в изопрен in vitro, в клеточном экстракте или in vivo. В репрезентативном анализе, клеточные экстракты получают путем культивирования штамма (например, описанного здесь штамма E.coli/pTrcKudzu) в шейкерной колбе методом, описанным в примере 1. После завершения индуцирования, приблизительно 10 мл клеток осаждают путем центрифугирования при 7000×g в течение 10 минут, и ресуспендируют в 5 мл PEB без глицерина. Клетки подвергают лизису в камере френч-пресса в соответствии со стандартными процедурами. Альтернативно, после процедуры замораживания/оттаивания при -80°С, клетки обрабатывают лизоцимом (раствором лизоцима Ready-Lyse; EpiCentre).
Активность полипептида изопренсинтазы в клеточном экстракте может быть определена, например, как описано в публикации Silver et al., J. Biol. Chem. 270:13010-13016, 1995 и в работах, цитируемых в данной публикации, каждая из которых во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание анализов на активность полипептида изопренсинтазы. DMAPP (Sigma) выпаривают досуха в потоке азота и подвергают регидратации до концентрации 100 мМ в 100 мМ фосфатно-калиевом буфере, pH 8,2, а затем помещают на хранение при -20°С. Для проведения этого анализа, раствор 5 мкл 1M МgCl2, 1 мМ (250 мкг/мл) DMAPP, 65 мкл буфера с растительным экстрактом (PEB) (50 мМ трис-HCl, pH 8,0, 20 мМ МgCl2, 5% глицерола и 2 мМ DTT) добавляют к 25 мкл клеточного экстракта в 20-миллилитровом сосуде, имеющем зону «хэдспейс» и снабженном завинчивающейся металлической крышкой и кремниевой перегородкой с тефлоновым покрытием (Agilent Technologies), и культивируют при 37°С в течение 15 минут со встряхиванием. Затем реакцию гасят добавлением 200 мкл 250 мМ EDTA, и продукт оценивают с помощью ГХ/МС, как описано в примере 1, части II.
Репрезентативными нуклеиновыми кислотами изопренсинтазы являются нуклеиновые кислоты, кодирующие полипептид, фрагмент полипептида, пептид и гибридный полипептид, обладающий по меньшей мере одной активностью полипептида изопренсинтазы. Репрезентативными полипептидами и нуклеиновыми кислотами изопренсинтазы являются природные полипептиды и нуклеиновые кислоты, происходящие от любого из описанных здесь организмов-источников, а также мутантные полипептиды и нуклеиновые кислоты, происходящие от любого из описанных здесь организмов-источников.
В некоторых вариантах изобретения, полипептид или нуклеиновая кислота изопренсинтазы происходит от семейства Fabaceae, такого как подсемейство Faboideae. В некоторых вариантах изобретения, полипептидом или нуклеиновой кислотой изопренсинтазы являются природный полипептид или природная нуклеиновая кислота, происходящие от Pueraria montana (пуэрарии волосистой) (Sharkey et al., Plant Physiology 137: 700-712, 2005); Pueraria lobata; тополя (такого как Populus alba x tremula CAC35696) (Miller et al., Planta 213: 483-487, 2001); осины (такой как Populus tremuloides) (Silver et al., JBC 270(22): 13010-1316, 1995) или английского дуба (Quercus robur) (Zimmer et al., WO 98/02550), где указанные публикации во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание, относящееся к нуклеиновым кислотам изопренсинтазы и к экспрессии полипептидов изопренсинтазы. Подходящими изопренсинтазами являются, но не ограничиваются ими, изопренсинтазы, идентифицированные в базе данных GenBank под рег. NN AY341431, AY316691, AY279379, AJ457070 и AY182241, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, в частности в описание, относящееся к последовательностям нуклеиновых кислот и полипептидов изопренсинтазы. В некоторых вариантах изобретения, полипептидом или нуклеиновой кислотой изопренсинтазы не является природный полипептид или природная нуклеиновая кислота, происходящие от Quercus robur (то есть указанным полипептидом или указанной нуклеиновой кислотой изопренсинтазы являются полипептид или нуклеиновая кислота изопренсинтазы, отличающиеся от природного полипептида или природной нуклеиновой кислоты, происходящих от Quercus robur). В некоторых вариантах изобретения, нуклеиновой кислотой или полипептидом изопренсинтазы не являются природный полипептид или природная нуклеиновая кислота, происходящие от тополя (такого как Populus alba x tremula CAC35696).
Репрезентативные полипептиды и нуклеиновые кислоты DXS
Как указывалось выше, полипептиды 1-дезокси-D-ксилулозо-5-фосфатсинтазы (DXS) превращают пируват и D-глицеральдегид-3-фосфат в 1-дезокси-D-ксилулозо-5-фосфат. Репрезентативными полипептидами DXS являются полипептиды, фрагменты полипептидов, пептиды и гибридные полипептиды, обладающие по меньшей мере одной активностью полипептида DXS. Для того, чтобы определить, обладает ли полипептид активностью полипептида DXS, могут быть применены стандартные методы (такие как методы, описанные в настоящей заявке), проводимые путем оценки способности полипептида превращать пируват и D-глицеральдегид-3-фосфат в 1-дезокси-D-ксилулозо-5-фосфат in vitro в клеточном экстракте или in vivo. Репрезентативными нуклеиновыми кислотами DXS являются нуклеиновые кислоты, кодирующие полипептид, фрагмент полипептида, пептид и гибридный полипептид, обладающий по меньшей мере одной активностью полипептида DXS. Репрезентативными полипептидами и нуклеиновыми кислотами DXS являются природные полипептиды и нуклеиновые кислоты, происходящие от любого из описанных здесь организмов-источников, а также мутантные полипептиды и нуклеиновые кислоты, происходящие от любого из описанных здесь организмов-источников.
Репрезентативные полипептиды и нуклеиновые кислоты IDI
Полипептиды изопентенилдифосфат-изомеразы (изопентенил-дифосфат-дельта-изомераза или IDI) катализируют взаимное превращение изопентенилдифосфата (IPP) и диметилаллилдифосфата (DMAPP) (например, превращение IPP в DMAPP и/или превращение DMAPP в IPP). Репрезентативными полипептидами IDI являются полипептиды, фрагменты полипептидов, пептиды и гибридные полипептиды, обладающие по меньшей мере одной активностью полипептида IDI. Для того, чтобы определить, обладает ли полипептид активностью полипептида IDI, могут быть применены стандартные методы (такие как методы, описанные в настоящей заявке), проводимые путем оценки способности данного полипептида к превращению IPP и DMAPP друг в друга in vitro, в клеточном экстракте или in vivo. Репрезентативными нуклеиновыми кислотами IDI являются нуклеиновые кислоты, кодирующие полипептид, фрагмент полипептида, пептид или гибридный полипептид, обладающий по меньшей мере одной активностью полипептида IDI. Репрезентативными полипептидами и нуклеиновыми кислотами IDI являются природные полипептиды и нуклеиновые кислоты, происходящие от любого из описанных здесь организмов-источников, а также мутантные полипептиды и нуклеиновые кислоты, происходящие от любого из описанных здесь организмов-источников.
Репрезентативные полипептиды и нуклеиновые кислоты пути MVA
Репрезентативными полипептидами пути MVA являются полипептиды ацетил-CoA-ацетилтрансферазы (AA-CoA-тиолазы), полипептиды 3-гидрокси-3-метилглутарил-CoA-синтазы (HMG-CoA-синтазы), полипептиды 3-гидрокси-3-метилглутарил-CoA-редуктазы (HMG-CoA-редуктазы), полипептиды мевалонаткиназы (MVK), полипептиды фосфомевалонаткиназы (PMK), полипептиды дифосфомевалонатдекарбоксилазы (MVD), полипептиды IDI и полипептиды (например, гибридные полипептиды), обладающие активностью двух или более полипептидов пути MVA. В частности, полипептидами пути MVA являются полипептиды, фрагменты полипептидов, пептиды и гибридные полипептиды, обладающие по меньшей мере одной активностью полипептида пути MVA. Репрезентативными нуклеиновыми кислотами пути MVA являются нуклеиновые кислоты, кодирующие полипептид, фрагмент полипептида, пептид и гибридный полипептид, обладающий по меньшей мере одной активностью полипептида пути MVA. Репрезентативными полипептидами и нуклеиновыми кислотами пути MVA являются природные полипептиды и нуклеиновые кислоты, происходящие от любого из описанных здесь организмов-источников, а также мутантные полипептиды и нуклеиновые кислоты, происходящие от любого из описанных здесь организмов-источников.
В частности, полипептиды ацетил-CoA-ацетилтрансферазы (AA-CoA-тиолазы или ААСТ) превращают две молекулы ацетил-CoA в ацетоацетил-CoA. Для того, чтобы определить, обладает ли полипептид активностью полипептида AA-CoA-тиолазы, могут быть применены стандартные методы (такие как методы, описанные в настоящей заявке), проводимые путем оценки способности данного полипептида превращать две молекулы ацетил-CoA в ацетоацетил-CoA in vitro, в клеточном экстракте или in vivo.
Полипептиды 3-гидрокси-3-метилглутарил-CoA-синтазы (HMG-CoA-синтазы или HMGS) превращают ацетоацетил-CoA в 3-гидрокси-3-метилглутарил-CoA. Для того, чтобы определить, обладает ли полипептид активностью полипептида HMG-CoA-синтазы, могут быть применены стандартные методы (такие как методы, описанные в настоящей заявке), проводимые путем оценки способности полипептида превращать ацетоацетил-CoA в 3-гидрокси-3-метилглутарил-CoA in vitro, в клеточном экстракте или in vivo.
Полипептиды 3-гидрокси-3-метилглутарил-CoA-редуктазы (HMG-CoA-редуктазы или HMGR) превращают 3-гидрокси-3-метилглутарил-CoA в мевалонат. Для того, чтобы определить, обладает ли полипептид активностью полипептида HMG-CoA-редуктазы, могут быть применены стандартные методы (такие как методы, описанные в настоящей заявке), проводимые путем оценки способности данного полипептида превращать 3-гидрокси-3-метилглутарил-CoA в мевалонат in vitro, в клеточном экстракте или in vivo.
Полипептиды мевалонат-киназы (MVK) фосфорилируют мевалонат с образованием мевалонат-5-фосфата. Для того, чтобы определить, обладает ли полипептид активностью полипептида MVK, могут быть применены стандартные методы (такие как методы, описанные в настоящей заявке), проводимые путем оценки способности данного полипептида превращать мевалонат в мевалонат-5-фосфат in vitro, в клеточном экстракте или in vivo.
Полипептиды фосфомевалонат-киназы (РMK) фосфорилируют мевалонат-5-фосфат с образованием мевалонат-5-дифосфата. Для того, чтобы определить, обладает ли полипептид активностью полипептида РMK, могут быть применены стандартные методы (такие как методы, описанные в настоящей заявке), проводимые путем оценки способности данного полипептида превращать мевалонат-5-фосфат в мевалонат-5-дифосфат in vitro, в клеточном экстракте или in vivo.
Полипептиды дифосфомевалонат-декарбоксилазы (MVD или DPMDC) превращают мевалонат-5-дифосфат в полипептиды изопентенил-дифосфата (IРР). Для того, чтобы определить, обладает ли полипептид активностью полипептида MVD, могут быть применены стандартные методы (такие как методы, описанные в настоящей заявке), проводимые путем оценки способности данного полипептида превращать мевалонат-5-дифосфат в IРР in vitro, в клеточном экстракте или in vivo.
Репрезентативные полипептиды и нуклеиновые кислоты IDI описаны выше.
Репрезентативные методы выделения нуклеиновых кислот
Нуклеиновые кислоты изопренсинтазы, DXS, IDI и/или пути MVA могут быть выделены стандартными методами. Методы получения нужных нуклеиновых кислот из представляющего интерес организма-источника (такого как бактериальный геном) широко применяются и хорошо известны специалистам в области молекулярной биологии (см., например, заявку WO 2004/033646 и цитируемые там работы, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание, относящееся к выделению представляющих интерес нуклеиновых кислот). Так, например, если последовательность нуклеиновой кислоты является известной (такой как любая из описанных здесь известных нуклеиновых кислот), то подходящие геномные библиотеки могут быть созданы путем расщепления рестриктирующей эндонуклеазой и могут быть скринированы с использованием зондов, комплементарных нужной последовательности нуклеиновой кислоты. После выделения последовательности, ДНК может быть амплифицирована с применением прямых методов амплификации с использованием стандартных праймеров, например, таких методов как полимеразная цепная реакция (ПЦР) (патент США № 4683202, который во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание, относящееся к ПЦР-методам) с получением ДНК в количестве, подходящем для ее трансформации с использованием соответствующих векторов.
Альтернативно, нуклеиновые кислоты изопренсинтазы, DXS, IDI, и/или пути MVA (такие как любые нуклеиновые кислоты изопренсинтазы, DXS, IDI, и/или пути MVA с известными нуклеотидными последовательностями) могут быть химически синтезированы стандартными методами.
Другие полипептиды и нуклеиновые кислоты изопренсинтазы, DXS, IDI или пути MVA, которые могут быть подходящими для их применения в описанных здесь композициях и способах, могут быть идентифицированы стандартными методами. Так, например, космидные библиотеки хромосомной ДНК организмов, которые, как известно, продуцируют изопрен, обычно конструируют в таких организмах, как E.coli, а затем скринируют на продуцирование изопрена. В частности, могут быть созданы космидные библиотеки, где крупные сегменты геномной ДНК (35-45 т.п.н.) упакованы в векторы и используются для трансформации соответствующих хозяев. Космидные векторы обладают уникальной способностью включать в себя большое количество ДНК. Обычно, космидные векторы имеют по меньшей мере одну копию последовательности космидной ДНК, которая необходима для упаковки и последующей циркуляризации гетерологичной ДНК. Эти векторы, помимо космидной последовательности, также содержат ориджин репликации, такой как ColEI, и маркеры резистентности к лекарственным средствам, такие как нуклеиновая кислота, резистентная к ампициллину или неомицину. Методы использования космидных векторов для трансформации подходящих бактериальных хозяев хорошо описаны в руководстве Sambrook et al., Molecular Cloning: A Laboratory Manual, 2nd ed., Cold Spring Harbor, 1989, которое во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание, относящееся к методам трансформации.
Обычно для клонирования космид, гетерологичную ДНК выделяют с использованием соответствующих рестриктирующих эндонуклеаз и лигируют непосредственно рядом с cos-областью космидного вектора с использованием соответствующих лигаз. Космидные векторы, содержащие линеаризованную гетерологичную ДНК, затем подвергают реакции с ДНК-упаковывающим носителем, таким как бактериофаг. В процессе упаковки, cos-сайты расщепляют и гетерологичную ДНК упаковывают в головную часть бактериальной вирусной частицы. Затем эти частицы используют для трансфекции подходящих клеток-хозяев, таких как E.coli. После инжекции в клетку, гетерологичная ДНК подвергается циркуляризации благодаря липким концам космиды. Таким образом, крупные сегменты гетерологичной ДНК могут быть введены в клетки-хозяева и могут экспрессироваться в этих клетках.
Другие методы получения нуклеиновых кислот изопренсинтазы, DXS, IDI и/или пути MVA включают скрининг метагеномной библиотеки с помощью анализа (такого как описанный здесь анализ в зоне «хэдспейс») или с помощью ПЦР с использованием праймеров, направленных на нуклеотиды, кодирующие последовательность из консервативных аминокислот (например, по меньшей мере 3 консервативных аминокислоты). Консервативные аминокислоты могут быть идентифицированы путем выравнивания аминокислотных последовательностей известных полипептидов изопренсинтазы, DXS, IDI и/или пути MVA. Консервативные аминокислоты полипептидов изопренсинтазы могут быть идентифицированы исходя из выровненных последовательностей известных полипептидов изопренсинтазы. Природный организм, который, как было обнаружено, продуцирует изопрен, может быть очищен стандартными методами очистки белков (хорошо известными специалистам), а затем полученный очищенный полипептид может быть секвенирован стандартными методами. Другие методы можно найти в литературе (см., например, публикации Julsing et al., Applied. Microbiol. Biotechnol. 75: 1377-84, 2007; и Withers et al., Appl Environ Microbiol. 73:6277-83, 2007, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание, относящееся к идентификации нуклеиновых кислот, участвующих в синтезе изопрена).
Кроме того, для идентификации других полипептидов и нуклеиновых кислот DXS, IDI или пути MVA могут быть использованы стандартные программы выравнивания последовательностей и/или предсказания их структуры, которые осуществляют исходя из сходства их первичных и/или предсказанных вторичных полипептидных структур со структурами известных полипептидов и нуклеиновых кислот DXS, IDI или пути MVA. Для идентификации полипептидов и нуклеиновых кислот изопренсинтазы, DXS, IDI или пути MVA могут быть также использованы стандартные базы данных, такие как база данных swissprot-trembl (сайт в Интернете «expasy.org», Швейцарского института биоинформатики Swiss-Prot group CMU - 1 rue Michel Servet CH-1211 Geneva 4, Switzerland). Вторичная и/или третичная структуры полипептида изопренсинтазы, DXS, IDI или пути MVA могут быть предсказаны с использованием параметров по умолчанию, имеющихся в стандартных программах предсказания структуры, таких как PredictProtein (Rost et al., The PredictProtein (630 West, 168 Street, BB217, New York, N.Y. 10032, USA). Альтернативно, фактическая вторичная и/или третичная структура полипептида изопренсинтазы, DXS, IDI или пути MVA может быть определена стандартными методами. Дополнительные нуклеиновые кислоты изопренсинтазы, DXS, IDI или пути MVA могут быть также идентифицированы путем гибридизации с зондами, генерируемыми из известных нуклеиновых кислот изопренсинтазы, DXS, IDI или пути MVA.
Репрезентативные промоторы и векторы
Любые описанные здесь нуклеиновые кислоты изопренсинтазы, DXS, IDI или пути MVA могут быть включены в один или более векторов. В соответствии с этим, настоящее изобретение также относится к векторам, содержащим одну или более нуклеиновых кислот, кодирующих любой из описанных здесь полипептидов изопренсинтазы, DXS, IDI или пути MVA. Используемый здесь термин «вектор» означает конструкцию, которая способна доставлять, а желательно, экспрессировать одну или более представляющих интерес нуклеиновых кислот в клетке-хозяине. Примерами таких векторов являются, но не ограничиваются ими, плазмиды, вирусные векторы, ДНК- или РНК-экспрессирующие векторы, космиды и фаговые векторы. В некоторых вариантах изобретения, вектор содержит нуклеиновую кислоту под контролем последовательности регуляции экспрессии.
Используемый здесь термин «последовательность регуляции экспрессии» означает последовательность нуклеиновой кислоты, которая направляет транскрипцию представляющей интерес нуклеиновой кислоты. Последовательностью регуляции экспрессии может быть промотор, такой как конститутивный или индуцибельный промотор, или энхансер. «Индуцибельный промотор» представляет собой промотор, который является активным при соответствующей регуляции условий окружающей среды или роста. Такая последовательность регуляции экспрессии функционально присоединена к транскрибируемому сегменту нуклеиновой кислоты.
В некоторых вариантах изобретения, указанный вектор содержит селективный маркер. Термин «селективный маркер» означает нуклеиновую кислоту, способную экспрессироваться в клетке-хозяине, что позволяет легко проводить отбор клеток-хозяев, содержащих введенную нуклеиновую кислоту или введенный вектор. Примерами селективных маркеров являются, но не ограничиваются ими, нуклеиновые кислоты резистентности к антибиотикам (например, к канамицину, ампициллину, карбенициллину, гентамицину, гигромицину, флеомицину, блеомицину, неомицину или хлорамфениколу) и/или нуклеиновые кислоты, сообщающие клеткам-хозяевам метаболические свойства, такие как преимущество в отношении питательных веществ. Репрезентативными селективными маркерами питательных веществ являются маркеры, известные специалистам, такие как amdS, argB и pyr4. Маркеры, используемые в векторных системах для трансформации Trichoderma, известны специалистам (см., например, публикации Finkelstein, Chapter 6 in Biotechnology of Filamentous Fungi, Finkelstein et al., Eds. Butterworth-Heinemann, Boston, MA, Chap. 6., 1992; и Kinghorn et al., Applied Molecular Genetics of Filamentous Fungi, Blackie Academic and Professional, Chapman and Hall, London, 1992, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание, относящееся к селективным маркерам). В некоторых вариантах изобретения, селективным маркером является нуклеиновая кислота amdS, которая кодирует фермент ацетамидазу, и позволяет трансформированным клеткам расти на ацетамиде как источнике азота. Применение нуклеиновой кислоты amdS A. nidulans в качестве селективного маркера описано в публикациях Kelley et al., EMBO J. 4:475-479, 1985 и Penttila et al., Gene 61:155-164, 1987 (которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание, относящееся к селективным маркерам). В некоторых вариантах изобретения, нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA интегрируется в хромосому клеток без селективного маркера.
Подходящими векторами являются векторы, совместимые с используемыми клетками-хозяевами. Подходящие векторы могут происходить, например, от бактерии, вируса (такого как бактериофаг T7 или фаг, происходящий от M-13), космиды, дрожжей или растения. Протоколы получения и применения таких векторов известны специалистам (см., например, руководство Sambrook et al., Molecular Cloning: A Laboratory Manual, 2nd ed., Cold Spring Harbor, 1989, которое во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание, относящееся к применению векторов).
Промоторы хорошо известны специалистам. Любой промотор, который функционирует в клетке-хозяине, может быть использован для экспрессии нуклеиновой кислоты изопренсинтазы, DXS, IDI или пути MVA в клетке-хозяине. Существует множество известных специалистам инициирующих экспрессию регуляторных областей или промоторов, которые могут быть использованы для запуска экспрессии нуклеиновых кислот изопренсинтазы, DXS, IDI или пути MVA в различных клетках-хозяевах (см., например, заявку WO 2004/033646 и цитируемые там работы, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание, относящееся к векторам, экспрессирующим представляющие интерес нуклеиновые кислоты). Фактически, в настоящем изобретении могут быть использованы любые промоторы, способные запускать экспрессию этих нуклеиновых кислот, включая, но не ограничиваясь ими, CYC1, HIS3, GAL1, GAL10, ADH1, PGK, PHO5, GAPDH, ADCI, TRP1, URA3, LEU2, ENO и TPI (используемые для экспрессии в Saccharomyces); AOX1 (используемые для экспрессии в Pichia); и lac, trp, λPL, λPR, T7, tac и trc (используемые для экспрессии в E.coli).
В некоторых вариантах изобретения используется промотор глюкозо-изомеразы (см., например, патент США No. 7132527 и цитируемые там работы, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание, относящееся к промоторам и плазмидным системам для экспрессии представляющих интерес полипептидов). Описанные мутанты промоторов глюкозо-изомеразы могут быть использованы для изменения уровней экспрессии полипептида, кодируемого нуклеиновой кислотой, функционально присоединенной к промотору глюкозо-изомеразы (патент США № 7132527). В различных вариантах изобретения, промотор глюкозо-изомеразы содержится в низкокопийных, среднекопийных или в высококопийных плазмидах (патент США № 7132527).
В различных вариантах изобретения, нуклеиновая кислота изопренсинтазы, DXS, IDI и/или пути MVA содержится в низкокопийной плазмиде (например, в плазмиде, которая включает по меньшей мере примерно 4 копии на клетку), в среднекопийной плазмиде (например, в плазмиде, которая содержит примерно от 10 до 15 копий на клетку) или в высококопийной плазмиде (например, в плазмиде, которая включает примерно 50 или более копий на клетку). В некоторых вариантах изобретения, гетерологичная или дополнительная эндогенная нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA функционально присоединена к промотору T7. В некоторых вариантах изобретения, гетерологичная или дополнительная эндогенная нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA, функционально присоединенная к промотору T7, содержится в среднекопийной или в высококопийной плазмиде. В некоторых вариантах изобретения, гетерологичная или дополнительная эндогенная нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA функционально присоединена к промотору Trc. В некоторых вариантах изобретения, гетерологичная или дополнительная эндогенная нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA, функционально присоединенная к промотору Trc, содержится в среднекопийной или в высококопийной плазмиде. В некоторых вариантах изобретения, гетерологичная или дополнительная эндогенная нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA функционально присоединена к промотору Lac. В некоторых вариантах изобретения, гетерологичная или дополнительная эндогенная нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA, функционально присоединенная к промотору Lac, содержится в низкокопийной плазмиде. В некоторых вариантах изобретения, гетерологичная или дополнительная эндогенная нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA функционально присоединена к эндогенному промотору, такому как эндогенный промотор Escherichia, Panteoa, Bacillus, Yarrowia, Streptomyces или Trichoderma, или к эндогенному промотору щелочной сериновой протеазы, изопренсинтазы, DXS, IDI или пути MVA. В некоторых вариантах изобретения, гетерологичная или дополнительная эндогенная нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA, функционально присоединенная к эндогенному промотору, содержится в высококопийной плазмиде. В некоторых вариантах изобретения, вектором является реплицирующаяся плазмида, которая не интегрируется в хромосому клеток. В некоторых вариантах изобретения, часть вектора или весь вектор интегрируется в хромосому клеток.
В некоторых вариантах изобретения, указанным вектором является вектор, который, при его введении в грибную клетку-хозяина, интегрируется в геном этой клетки-хозяина и реплицируется в ней. Список векторов приводится в каталоге штаммов: Fungal Genetics Stock Center Catalogue of Strains (FGSC, www на сайте «fgsc.net», и в цитируемых там работах, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание векторов). Другие примеры подходящих экспрессионных векторов и/или интегрирующихся векторов приводятся в руководствах Sambrook et al., Molecular Cloning: A Laboratory Manual, 2nd ed., Cold Spring Harbor, 1989, Current Protocols in Molecular Biology (F. M. Ausubel et al. (eds) 1987, Supplement 30, section 7.7.18); van den HoNdeI et al. in Bennett and Lasure (Eds.) More Gene Manipulations in Fungi, Academic Press pp. 396-428, 1991; и в патенте США № 5874276, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание векторов. Особенно подходящими векторами являются pFB6, pBR322, PUC18, pUC100 и pENTR/D.
В некоторых вариантах изобретения, нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA функционально присоединена к подходящему промотору, который обладает транскрипционной активностью в клетках-хозяевах грибов. Такой промотор может происходить от одной или более нуклеиновых кислот, кодирующих полипептид, который является эндогенным или гетерологичным для клетки-хозяина. В некоторых вариантах изобретения, промотор может быть использован в хозяине Trichoderma. Подходящими неограничивающими примерами промоторов являются cbh1, cbh2, egl1, egl2, pepA, hfb1, hfb2, xyn1 и amy. В некоторых вариантах изобретения, указанным промотором является промотор, который является нативным для клетки-хозяина. Так, например, в некоторых вариантах изобретения, если хозяином является T. reesei, то промотором является нативный промотор T. reesei. В некоторых вариантах изобретения, промотором является промотор cbh1 T. reesei, который представляет собой индуцибельный промотор, депонированный в GenBank под рег. № D86235, который во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание, относящееся к промоторам. В некоторых вариантах изобретения, указанный промотор представляет собой промотор, который является гетерологичным для данной грибной клетки-хозяина. Другими примерами подходящих промоторов являются промоторы генов глюкоамилазы (glaA) A. awamori и A. niger (публикации Nunberg et al., Mol. Cell Biol. 4:2306-2315, 1984 и Boel et al., EMBO J. 3:1581-1585, 1984, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание промоторов); альфа-амилазы Aspergillus niger, TAKA-амилазы Aspergillus oryzae, xln1 T. reesei и целлобиогидролазы 1 T. reesei (EP 137280, который во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание промоторов).
В некоторых вариантах изобретения, экспрессионный вектор также включает последовательность терминации. Регуляторные области терминации транскрипции могут также происходить от различных генов, которые являются нативными для клеток-хозяев. В некоторых вариантах изобретения, последовательность терминации и промоторная последовательность происходят от одного и того же источника. В другом варианте изобретения, последовательность терминации является эндогенной для клетки-хозяина. Особенно подходящей последовательностью-терминатором является cbh1, происходящая от штамма Trichoderma (такого как T. reesei). Другие подходящие терминаторы, происходящие от грибов, включают терминатор, происходящий от нуклеиновой кислоты глюкоамилазы A. niger или A. awamori (публикации Nunberg et al., Mol. Cell Biol. 4:2306-2315, 1984 и Boel et al., EMBO J. 3:1581-1585, 1984, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание терминаторов, происходящих от грибов). Может быть включен, но необязательно, сайт терминации. Для эффективной экспрессии полипептидов, ДНК, кодирующую указанный полипептид, функционально присоединяют посредством инициирующих кодонов к выбранным областям регуляции экспрессии, так, чтобы такая экспрессия приводила к образованию соответствующей матричной РНК.
В некоторых вариантах изобретения, промотор, кодирующая область и терминатор происходят от экспрессируемой нуклеиновой кислоты изопренсинтазы, DXS, IDI или пути MVA. В некоторых вариантах изобретения, кодирующую область нуклеиновой кислоты изопренсинтазы, DXS, IDI или пути MVA встраивают в многоцелевой экспрессионный вектор так, чтобы он находился под транскрипционным контролем последовательности экспрессионной промоторной конструкции и последовательности терминатора. В некоторых вариантах изобретения, гены или их часть встраивают в область, расположенную ниже сильного промотора cbh1.
Нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA может быть встроена в вектор, такой как экспрессионный вектор, с применением стандартных методов (см. руководство Sambrook et al., Molecular Cloning: A Laboratory Manual, Cold Spring Harbor, 1982, которое во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание, относящееся к скринингу соответствующих последовательностей ДНК и конструированию векторов). Методы, применяемые для лигирования конструкции ДНК, содержащей представляющую интерес нуклеиновую кислоту (такую как нуклеиновая кислота изопренсинтазы, DXS, IDI или пути MVA), промотор, терминатор и другие последовательности, и для их встраивания в подходящий вектор, хорошо известны специалистам. Так, например, рестриктирующие ферменты могут быть использованы для расщепления нуклеиновой кислоты изопренсинтазы, DXS, IDI или пути MVA, и вектора. Затем, совместимые концы расщепленной нуклеиновой кислоты изопренсинтазы, DXS, IDI или пути MVA и расщепленный вектор могут быть лигированы. Связывание обычно осуществляют путем лигирования в стандартных рестрикционных сайтах. Если такие сайты отсутствуют, то используют синтетические олигонуклеотидные линкеры в соответствии со стандартной практикой (см. руководства Sambrook et al., Molecular Cloning: A Laboratory Manual, 2nd ed., Cold Spring Harbor, 1989, и Bennett and Lasure, More Gene Manipulations in Fungi, Academic Press, San Diego, pp 70-76, 1991, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание, относящееся к олигонуклеотидным линкерам). Кроме того, векторы могут быть сконструированы с применением стандартных рекомбинантных методов (например, Invitrogen Life Technologies, Gateway Technology).
В некоторых вариантах изобретения, может оказаться желательной сверхэкспрессия нуклеиновых кислот изопренсинтазы, DXS, IDI или пути MVA на уровнях, которые значительно превышают уровни, обычно наблюдаемые в природных клетках. Такая сверхэкспрессия может быть осуществлена путем селективного клонирования нуклеиновых кислот, кодирующих указанные полипептиды в многокопийные плазмиды под контролем сильного индуцибельного или конститутивного промотора. Методы сверхэкспрессии желательных полипептидов широко применяются и хорошо известны специалистам в молекулярной биологии, и их описание можно найти в руководстве Sambrook et al., Molecular Cloning: A Laboratory Manual, 2nd ed., Cold Spring Harbor, 1989, которое во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, его в описание, относящееся к методам клонирования.
Нижеследующие материалы включают описание дополнительных общих методов, которые могут быть применены в настоящем изобретении, а именно, руководства Kreigler, Gene Transfer and Expression; A Laboratory Manual, 1990; и Ausubel et al., Eds. Current Protocols in Molecular Biology, 1994, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание, относящееся к методам молекулярной биологии и клонирования.
Репрезентативные организмы-источники
Нуклеиновые кислоты изопренсинтазы, DXS, IDI или пути MVA (и кодируемые ими полипептиды) могут быть получены из любого организма, который обычно содержит нуклеиновые кислоты изопренсинтазы, DXS, IDI или пути MVA. Как указывалось выше, изопрен обычно продуцируется различными организмами, такими как бактерии, дрожжи, растения и животные. Такие организмы содержат полипептиды пути MVA, пути DXP или путей MVA и DXP, по которым продуцируется изопрен (фигура 19). Таким образом, нуклеиновые кислоты DXS могут быть получены, например, из любого организма, содержащего нуклеиновые кислоты пути DXP или путей MVA и DXP. Нуклеиновые кислоты IDI и изопренсинтазы могут быть получены, например, из любого организма, содержащего нуклеиновые кислоты пути MVA, пути DXP или тех и других путей. Нуклеиновые кислоты пути MVA могут быть получены, например, из любого организма, содержащего нуклеиновые кислоты пути MVA или путей MVA и DXP.
В некоторых вариантах изобретения, последовательность нуклеиновой кислоты изопренсинтазы, DXS, IDI или пути MVA идентична последовательности нуклеиновой кислоты, которая продуцируется любыми из нижеследующих организмов в природе. В некоторых вариантах изобретения, аминокислотная последовательность изопренсинтазы, DXS, IDI или пути MVA идентична полипептидной последовательности, которая продуцируется в природе любыми из нижеследующих организмов. В некоторых вариантах изобретения, нуклеиновой кислотой или полипептидом изопренсинтазы, DXS, IDI или пути MVA являются мутантная нуклеиновая кислота или мутантный полипептид, происходящие от любого из описанных здесь организмов. Используемый здесь термин «происходит от...» относится к источнику нуклеиновой кислоты или полипептида, в который были введены одна или более мутаций. Так, например, термин «полипептид, происходящий от растительного полипептида» означает представляющий интерес полипептид, который образуется в результате введения одной или более мутаций в последовательность растительного полипептида дикого типа (то есть в его природную последовательность).
В некоторых вариантах изобретения, организмом-источником являются грибы, примерами которых могут служить виды Aspergillus, такие как A. oryzae и A. niger, виды Saccharomyces, такие как S. cerevisiae, виды Schizosaccharomyces, такие как S. pombe, и виды Trichoderma, такие как T. reesei. В некоторых вариантах изобретения, организмом-источником являются клетки нитчатых грибов. Термин «нитчатые грибы» означает все нитчатые формы подразделения Eumycotina (см. Alexopoulos, C. J. (1962), Introductory Mycology, Wiley, New York). Эти грибы характеризуются наличием вегетативного мицелия, клеточная стенка которого состоит из хитина, целлюлозы и других сложных полисахаридов. Нитчатые грибы отличаются от дрожжей по своим морфологическим, физиологическим и генетическим свойствам. Вегетативный рост нитчатых грибов происходит за счет удлинения гифов, а катаболизм углерода в этих грибах является облигатно аэробным. Родительскими клетками нитчатых грибов могут быть клетки видов, которыми являются, но не ограничиваются ими, Trichoderma (например, Trichoderma reesei, вегетативная морфа грибов Hypocrea jecorina, ранее классифицированных как T. longibrachiatum, Trichoderma viride, Trichoderma koningii, Trichoderma harzianum) (Sheir-Neirs et al., Appl. Microbiol. Biotechnol 20: 46-53, 1984; ATCC No. 56765 и ATCC No. 26921); Penicillium sp., Humicola sp. (например, H. insolens, H. lanuginose или H. grisea); Chrysosporium sp. (например, C. lucknowense), Gliocladium sp., Aspergillus sp. (например, A. oryzae, A. niger, A sojae, A. japonicus, A. nidulans или A. awamori) (Ward et al., Appl. Microbiol. Biotechnol. 39: 7380743, 1993 и Goedegebuur et al., Genet 41: 89-98, 2002), Fusarium sp., (например, F. roseum, F. graminum F. cerealis, F. oxysporuim, или F. venenatum), Neurospora sp., (например, N. crassa), Hypocrea sp., Mucor sp., (например, M. miehei), Rhizopus sp. и Emericella sp. (см. также, Innis et al., Sci. 228: 21-26, 1985). Термины «Trichoderma» или «Trichoderma sp.» или «Trichoderma spp.» относятся к грибам любого рода, которые ранее или в настоящее время были классифицированы как Trichoderma.
В некоторых вариантах изобретения, такими грибами являются A. nidulans, A. awamori, A. oryzae, A. aculeatus, A. niger, A. japonicus, T. reesei, T. viride, F. oxysporum или F. solani. Штаммы Aspergillus описаны в публикациях Ward et al., Appl. Microbiol. Biotechnol. 39:738-743, 1993 и Goedegebuur et al., Curr Gene 41:89-98, 2002, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание грибов. В конкретных вариантах изобретения, грибами являются штаммы Trichoderma, такие как штамм T. reesei. Штаммы T. reesei известны специалистам и неограничивающими примерами таких штаммов являются штаммы ATCC No. 13631, ATCC No. 26921, ATCC No. 56764, ATCC No. 56765, ATCC No. 56767 и NRRL 15709, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание штаммов T. reesei. В некоторых вариантах изобретения, штаммом-хозяином является производное RL-P37. RL-P37 описан в публикации Sheir-Neiss et al., Appl. Microbiol. Biotechnology 20:46-53, 1984, которая во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание штаммов T. reesei.
В некоторых вариантах изобретения, организмом-источником являются дрожжи, такие как Saccharomyces sp., Schizosaccharomyces sp., Pichia sp., или Candida sp.
В некоторых вариантах изобретения, организмом-источником является бактерия, такая как штаммы Bacillus, такие как B. lichenformis или B. subtilis, штаммы Pantoea, такие как P. citrea, штаммы Pseudomonas, такие как P. alcaligenes, штаммы Streptomyces, такие как S. lividans или S. rubiginosus, или штаммы Escherichia, такие как E.coli.
Используемый здесь термин «род Bacillus» включает все виды, принадлежащие к роду «Bacillus», известному специалистам в данной области, включая, но не ограничиваясь ими, B. subtilis, B. licheniformis, B. lentus, B. brevis, B. stearothermophilus, B. alkalophilus, B. amyloliquefaciens, B. clausii, B. halodurans, B. megaterium, B. coagulans, B. circulans, B. lautus и B. thuringiensis. Следует отметить, что род Bacillus может подвергаться таксономической реорганизации. Таким образом, предполагается, что такой род включает виды, которые были переклассифицированы, включая, но не ограничиваясь ими, такие микроорганизмы, как B. stearothermophilus, который в настоящее время называется «Geobacillus stearothermophilus». Продуцирование резистентных эндоспор в присутствии кислорода рассматривается как определяющий признак рода Bacillus, хотя такими свойствами также обладают недавно идентифицированные бактерии AliCYC1obacillus, Amphibacillus, Aneurinibacillus, Anoxybacillus, Brevibacillus, Filobacillus, Gracilibacillus, Halobacillus, Paenibacillus, Salibacillus, Thermobacillus, Ureibacillus и Virgibacillus.
В некоторых вариантах изобретения, организмом-источником является грам-положительная бактерия. Неограничивающими примерами являются штаммы Streptomyces (например, S. lividans, S. coelicolor, или S. griseus) и Bacillus. В некоторых вариантах изобретения, организмом-источником является грам-отрицательная бактерия, такая как E.coli или Pseudomonas sp.
В некоторых вариантах изобретения, организмами-источниками являются растения, такие как растения семейства бобовых (Fabaceae), такие как растение подсемейства Faboideae. В некоторых вариантах изобретения, организмами-источниками являются пуэрария, тополь (такой как Populus alba x tremula CAC35696), осина (такая как Populus tremuloides), или Quercus robur.
В некоторых вариантах изобретения, организмами-источниками являются водоросли, такие как зеленые водоросли, красные водоросли, глаукофиты, хлорарахниофиты, эвглениды, хромиста, или динофлагелляты.
В некоторых вариантах изобретения, организмами-источниками являются цианобактерии, такие как цианобактерии, классифицированные по любой из нижеследующих групп на основе их морфологии: Chroococcales, Pleurocapsales, Oscillatoriales, Nostocales или Stigonematales.
Репрезентативные клетки-хозяева
Для экспрессии полипептидов изопренсинтазы, DXS, IDI и/или пути MVA и для продуцирования изопрена способами, заявленными в настоящем изобретении, могут быть использованы различные клетки-хозяева. Репрезентативными клетками-хозяевами являются клетки любого из организмов, перечисленных в предыдущем разделе под заголовком «Репрезентативные организмы-источники». Клеткой-хозяином может быть клетка, которая по своей природе продуцирует изопрен, или клетка, которая по своей природе не продуцирует изопрен. В некоторых вариантах изобретения, клетка-хозяин по своей природе продуцирует изопрен по пути DXP, а для повышения уровня продуцирования изопрена по этому пути добавляют нуклеиновую кислоту изопренсинтазы, DXS и/или IDI. В некоторых вариантах изобретения, клетка-хозяин по своей природе продуцирует изопрен по пути MVA, а для повышения уровня продуцирования изопрена по этому пути добавляют нуклеиновые кислоты изопренсинтазы и/или нуклеиновые кислоты одного или более путей MVA. В некоторых вариантах изобретения, клетка-хозяин по своей природе продуцирует изопрен по пути DXP, а для продуцирования изопрена по неполному или полному пути MVA, а также пути DXP добавляют одну или более нуклеиновых кислот пути MVA. В некоторых вариантах изобретения, клетка-хозяин по своей природе продуцирует изопрен по путям DXP и MVA, а для повышения уровня продуцирования изопрена по одному или обоим этим путям добавляют одну или более нуклеиновых кислот изопренсинтазы, DXS, IDI или пути MVA.
Репрезентативные методы трансформации
Нуклеиновые кислоты изопренсинтазы, DXS, IDI и/или пути MVA или содержащие их векторы могут быть встроены в клетку-хозяина (например, в описанные здесь клетки растений, клетки грибов, клетки дрожжей или в бактериальные клетки) стандартными методами экспрессии кодируемого полипептида изопренсинтазы, DXS, IDI и/или пути MVA. Введение ДНК-конструкции или ДНК-вектора в клетку-хозяина может быть осуществлено такими методами, как трансформация, электропорация, микроинжекция в ядро; трансдукция; трансфекция (например, трансфекция, опосредуемая липофекцией или DEAE-декстрином; или трансфекция с использованием рекомбинантного фагового вируса); инкубирование с преципитатом ДНК, содержащим фосфат кальция; высокоскоростная бомбардировка микрочастицами, покрытыми ДНК; и слияние протопластов. Общие методы трансформации известны специалистам (см., например, руководства Current Protocols in Molecular Biology (F. M. Ausubel et al. (eds) Chapter 9, 1987; Sambrook et al., Molecular Cloning: A Laboratory Manual, 2nd ed., Cold Spring Harbor, 1989; и Campbell et al., Curr Genet, 16:53-56, 1989, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание методов трансформации). Экспрессия гетерологичного полипептида в Trichoderma описана в патенте США No. 6022725; в патенте США No. 6268328; в патенте США No. 7262041; в заявке WO 2005/001036; Harkki et al; Enzyme Microb. Technol. 13:227-233, 1991; Harkki et al., Bio Technol. 7:596-603, 1989; EP 244234; EP 215594; и Nevalainen et al., "The Molecular Biology of Trichoderma and its Application to the Expression of Both Homologous and Heterologous Genes," in Molecular Industrial Mycology, Eds. Leong and Berka, Marcel Dekker Inc., NY pp. 129-148, 1992, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание методов трансформации и экспрессии). Описание трансформации штаммов Aspergillus можно также найти в публикациях Cao et al., (Sci. 9:991-1001, 2000; EP 238023; и Yelton et al., Proceedings. Natl. Acad. Sci USA 81:1470-1474, 1984 (которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание методов трансформации). Введенные нуклеиновые кислоты могут интегрироваться в хромосомную ДНК или сохраняться в виде внехромосомных реплицирующихся последовательностей.
Для отбора трансформантов может быть применен любой известный метод. В одном из неограничивающих примеров, стабильные трансформанты, включающие маркер amdS, отличаются от нестабильных трансформантов более высокой скоростью роста и образованием кольцевых колоний, имеющих гладкую, а не шероховатую поверхность на твердой культуральной среде, содержащей ацетамид. Кроме того, в некоторых случаях, дополнительный тест на стабильность проводят путем культивирования трансформантов на твердой неселективной среде (например, на среде, не содержащей ацетамида), сбора спор из этой культуральной среды, и определения процента спор, которые затем начинают прорастать и растут на селективной среде, содержащей ацетамид.
В некоторых вариантах изобретения, клетки грибов трансформируют способом, включающим образование протопластов и трансформацию протопластов с последующей регенерацией клеточной стенки известным методом. В одном из конкретных вариантов изобретения, получение Trichoderma sp. для трансформации включает получение протопластов из грибного мицелия (см. публикацию Campbell et al., Curr. Genet. 16:53-56, 1989, которая во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание методов трансформации). В некоторых вариантах изобретения, мицелий образуется из проросших вегетативных спор. Этот мицелий обрабатывают ферментом, который гидролизует клеточную стенку с образованием протопластов. Затем протопласты сохраняют путем включения в суспендирующую среду осмотического стабилизатора. Такими стабилизаторами являются сорбит, маннит, хлорид калия, сульфат магния и т.п. Обычно, концентрация этих стабилизаторов варьируется в пределах от 0,8 M до 1,2 M. При этом, желательно, чтобы суспендирующая среда содержала примерно 1,2 M раствора сорбита.
Поглощение ДНК штаммом-хозяином Trichoderma sp. зависит от концентрации ионов кальция. Обычно, раствор, используемый для поглощения, содержит примерно от 10 мM CaCl2 до 50 мM CaCl2. Раствор, используемый для поглощения, помимо ионов кальция обычно содержит и другие соединения, такие как забуферивающая система, такая как буфер TE (10 мM трис, pH 7,4; 1 мM EDTA) или 10 мМ буфера MOPS, pH 6,0 (морфолинпропансульфоновая кислота) и полиэтиленгликоль (ПЭГ). Не ограничиваясь какой-либо конкретной теорией, следует отметить, что полиэтиленгликоль, очевидно, связывается с клеточными мембранами, что позволяет содержимому среды поступать в цитоплазму штамма Trichoderma sp. и тем самым переносить плазмидную ДНК в ядро. Такой гибрид часто сохраняет множество копий плазмидной ДНК, интегрированной в хромосому хозяина.
Обычно, для трансформации используют суспензию, содержащую протопласты или клетки Trichoderma sp., которые были подвергнуты обработке для сообщения им проницаемости, при плотности 105-107/мл (например, 2×106/мл). Эти протопласты или клетки в объеме 100 мкл в соответствующем растворе (например, 1,2 M сорбита и 50 мM CaCl2) смешивают с нужной ДНК. Обычно, в раствор, используемый для поглощения, добавляют высокую концентрацию ПЭГ. В суспензию протопластов может быть добавлен 0,1-1 объем 25% ПЭГ 4000. В некоторых вариантах изобретения, в суспензию протопластов добавляют примерно 0,25 объема. В раствор для поглощения и для стимуляции трансформации могут быть также введены добавки, такие как диметилсульфоксид, гепарин, спермидин, хлорид калия и т.п. Аналогичные процедуры могут быть применены и к другим клеткам-хозяевам, происходящим от грибов (см., например, патенты США №№6022725 и 6268328, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание методов трансформации).
Затем смесь обычно культивируют приблизительно при 0°C в течение периода времени от 10 до 30 минут. Для усиления поглощения нужной последовательности нуклеиновой кислоты, к смеси добавляют дополнительное количество ПЭГ. 25% ПЭГ4000 обычно добавляют в объеме, который в 5-15 раз превышает объем смеси для трансформации, однако, могут быть добавлены большие и меньшие объемы. Желательно, чтобы объем 25% ПЭГ4000 приблизительно в 10 раз превышал объем смеси для трансформации. После добавления ПЭГ, смесь для трансформации культивируют либо при комнатной температуре, либо на льду, а затем добавляют раствор сорбита и CaCl2. Затем к расплавленным аликвотам культуральной среды добавляют суспензию протопластов. Если культуральная среда включает вещества для отбора на рост клеток (например, ацетамид или антибиотики), то это позволяет выявлять только рост трансформантов.
Трансформация бактериальных клеток может быть осуществлена стандартными методами, например, как описано в руководстве Sambrook et al., Molecular Cloning: A Laboratory Manual, Cold Spring Harbor, 1982, которое во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание методов трансформации.
Репрезентативные среды для культивирования клеток
Настоящее изобретение также включает клетки или популяции клеток в культуре, которые продуцируют изопрен. Термин «клетки в культуре» означает две или более клеток в растворе (например, в клеточной среде), в котором эти клетки могут подвергаться одному или более клеточным делениям. Термин «клетки в культуре» не включает растительные клетки, которые являются частью «живых» многоклеточных растений, содержащих клетки, дифференцирующиеся в растительные ткани. В различных вариантах изобретения, клеточная культура включает по меньшей мере или примерно 10, 20, 50, 100, 200, 500, 1000, 5000, 10000 или более клеток.
Для культивирования клеток-хозяев может быть использован любой источник углерода. Термин «источник углерода» означает одно или более углерод-содержащих соединений, способных подвергаться метаболизму в клетке- или организме-хозяине. Так, например, клеточная среда, используемая для культивирования клеток-хозяев, может включать любой источник углерода, подходящий для поддержания жизнеспособности или роста клеток-хозяев.
В некоторых вариантах изобретения, источником углерода является углевод (такой как моносахарид, дисахарид, олигосахарид или полисахариды), инвертированный сахар (например, ферментативно обработанный сахарный сироп), глицерол, глицерин (например, глицериновый побочный продукт, образующийся в результате производства биодизельного топлива или мыла), дигидроксиацетон, одноатомный источник углерода, масло, (например, масло, полученное из целого растения или из его семян (например, кукурузное масло, пальмовое масло или соевое масло), животный жир, масло животного происхождения, жирная кислота (например, насыщенная жирная кислота, ненасыщенная жирная кислота или полиненасыщенная жирная кислота), липид, фосфолипид, глицеролипид, моноглицерид, диглицерид, триглицерид, полипептид (например, микробный или растительный белок или пептид), возобновляемый источник углерода (например, источник углерода, представляющий собой биомассу, такую как гидролизованная биомасса); дрожжевой экстракт, компонент дрожжевого экстракта, полимер, кислота, спирт, альдегид, кетон, аминокислота, сукцинат, лактат, ацетат, этанол или любая комбинация двух или более вышеперечисленных компонентов. В некоторых вариантах изобретения, источником углерода является продукт фотосинтеза, включая, но не ограничиваясь им, глюкозу.
Репрезентативными моносахаридами являются глюкоза и фруктоза; репрезентативными олигосахаридами являются лактоза и сахароза, а репрезентативными полисахаридами являются крахмал и целлюлоза. Репрезентативными углеводами являются C6-сахара (например, фруктоза, манноза, галактоза или глюкоза) и C5-сахара (например, ксилоза или арабиноза). В некоторых вариантах изобретения, клеточная среда включает углевод, а также источник углерода, не являющийся углеводом (например, глицерол, глицерин, дигидроксиацетон, одноатомный источник углерода, масло, животный жир, масло животного происхождения, жирная кислота, липид, фосфолипид, глицеролипид, моноглицерид, диглицерид, триглицерид, возобновляемый источник углерода или компонент дрожжевого экстракта). В некоторых вариантах изобретения, клеточная среда включает углевод, а также полипептид (например, микробный или растительный белок или пептид). В некоторых вариантах изобретения, микробным полипептидом является полипептид дрожжей или бактерий. В некоторых вариантах изобретения, растительным полипептидом является полипептид, происходящий от сои, кукурузы, канолы, ятропы, пальмы, арахиса, подсолнечника, кокосового ореха, горчицы, рапса, семян хлопчатника, косточек плодов пальмы, плодов маслины, сафлора, кунжута или семян льна.
В некоторых вариантах изобретения, концентрация углевода составляет по меньшей мере или примерно 5 граммов на литр бульона (г/л, где указанный объем бульона включает объем клеточной среды и объем клеток), например, по меньшей мере или примерно 10, 15, 20, 30, 40, 50, 60, 80, 100, 150, 200, 300, 400 г/л или более. В некоторых вариантах изобретения, концентрация углевода составляет примерно от 50 до 400 г/л, например, примерно от 100 до 360 г/л, примерно от 120 до 360 г/л или примерно от 200 до 300 г/л. В некоторых вариантах изобретения, эта концентрация углевода включает общее количество углевода, добавляемого до и/или во время культивирования клеток-хозяев.
В некоторых вариантах изобретения, клетки культивируют в условиях с ограниченным содержанием глюкозы. Термин «условия с ограниченным содержанием глюкозы» означает, что количество добавленной глюкозы составляет менее чем или примерно 105% (например, 100%) от количества глюкозы, потребляемой клетками. В конкретных вариантах изобретения, количество глюкозы, добавляемой в культуральную среду, приблизительно равно количеству глюкозы, потребляемой клетками в течение определенного периода времени. В некоторых вариантах изобретения, скорость клеточного роста регулируется путем ограничения количества добавляемой глюкозы, так, чтобы клетки могли расти на уровне, который может поддерживаться определенным количеством глюкозы в клеточной среде. В некоторых вариантах изобретения, глюкоза не накапливается в процессе культивирования клеток. В различных вариантах изобретения, клетки культивируют в условиях с ограниченным содержанием глюкозы в течение более чем или примерно 1, 2, 3, 5, 10, 15, 20, 25, 30, 35, 40, 50, 60, или 70 часов. В различных вариантах изобретения, клетки культивируют в условиях с ограниченным содержанием глюкозы в течение более чем или примерно 5, 10, 15, 20, 25, 30, 35, 40, 50, 60, 70, 80, 90, 95, или 100% от общего периода времени культивирования клеток. Не ограничиваясь какой-либо конкретной теорией, следует отметить, что такие условия с ограниченным содержанием глюкозы, очевидно, являются более предпочтительными для регуляции роста клеток.
В некоторых вариантах изобретения, клетки культивируют в присутствии избыточного количества глюкозы. В конкретных вариантах изобретения, количество добавляемой глюкозы примерно более чем на 105% (например, примерно или более чем 110, 120, 150, 175, 200, 250, 300, 400 или 500%) превышает количество глюкозы, потребляемое клетками в конкретный период времени. В некоторых вариантах изобретения, глюкоза накапливается в процессе культивирования клеток.
Репрезентативными липидами являются любые вещества, содержащие одну или более жирных кислот, которые представляют собой жирные кислоты, имеющие 4 или более атомов углерода и которые являются насыщенными, ненасыщенными или разветвленными.
Репрезентативными маслами являются липиды, которые имеют жидкую форму при комнатной температуре. В некоторых вариантах изобретения, липиды содержат одну или более жирных кислот с 4 атомами углерода или более (например, они содержат одну или более насыщенных, ненасыщенных или разветвленных жирных кислот с четырьмя или более атомами углерода). В некоторых вариантах изобретения, масло получают из сои, кукурузы, канолы, ятрофы, пальмы, арахиса, подсолнечника, кокосового ореха, горчицы, рапса, семян хлопчатника, косточек плодов пальмы, плодов маслины, сафлора, кунжута, семян льна, масло-содержащих микробных клеток, китайского жира или любых комбинаций двух или более масел вышеуказанных материалов.
Репрезентативными жирными кислотами являются соединения формулы R-COOH, где «R» представляет собой углеводород. Репрезентативными ненасыщенными жирными кислотами являются соединения, в которых «R» включает по меньшей мере одну углерод-углеродную двойную связь. Репрезентативными ненасыщенными жирными кислотами являются, но не ограничиваются ими, олеиновая кислота, вакценовая кислота, линолевая кислота, пальмитолеиновая кислота и арахидоновая кислота. Репрезентативными полиненасыщенными жирными кислотами являются соединения, в которых «R» включает множество углерод-углеродных двойных связей. Репрезентативными насыщенными жирными кислотами являются соединения, в которых «R» представляет собой насыщенную алифатическую группу. В некоторых вариантах изобретения, источник углерода включает одну или более С12-С22-жирных кислот, таких как С12-насыщенная жирная кислота, С14-насыщенная жирная кислота, С16-насыщенная жирная кислота, С18-насыщенная жирная кислота, С20-насыщенная жирная кислота или С22-насыщенная жирная кислота. В репрезентативном варианте изобретения, жирной кислотой является пальмитиновая кислота. В некоторых вариантах изобретения, источником углерода является соль жирной кислоты (например, ненасыщенной жирной кислоты), производное жирной кислоты (например, ненасыщенной жирной кислоты) или соль производного жирной кислоты (например, ненасыщенной жирной кислоты). Подходящими солями являются, но не ограничиваются ими, соли лития, соли калия, соли натрия и т.п. Ди- и триглицеролы представляют собой сложные эфиры глицерола и жирных кислот.
В некоторых вариантах изобретения, концентрация липида, масла, жира, жирной кислоты, моноглицерида, диглицерида или триглицерида составляет по меньшей мере или примерно 1 грамм на литр бульона (г/л, где указанный объем бульона включает объем клеточной среды и объем клеток), например, по меньшей мере или примерно 5, 10, 15, 20, 30, 40, 50, 60, 80, 100, 150, 200, 300, 400 г/л или более. В некоторых вариантах изобретения, концентрация липида, масла, жира, жирной кислоты, моноглицерида, диглицерида или триглицерида составляет примерно от 10 до 400 г/л, например, примерно от 25 до 300 г/л, примерно от 60 до 180 г/л или примерно от 75 до 150 г/л. В некоторых вариантах изобретения, указанная концентрация включает общее количество липида, масла, жира, жирной кислоты, моноглицерида, диглицерида или триглицерида, добавляемое до и/или во время культивирования клеток-хозяев. В некоторых вариантах изобретения, источник углерода включает (i) липид, масло, жир, жирную кислоту, моноглицерид, диглицерид или триглицерид и (ii) углевод, такой как глюкоза. В некоторых вариантах изобретения, отношение количества липида, масла, жира, жирной кислоты, моноглицерида, диглицерида или триглицерида к количеству углевода составляет примерно 1:1 в расчете на атом углерода (то есть один атом углерода в липиде, масле, жире, жирной кислоте, моноглицериде, диглицериде или триглицериде на один атом углерода в углеводе). В конкретных вариантах изобретения, количество липида, масла, жира, жирной кислоты, моноглицерида, диглицерида или триглицерида составляет примерно от 60 до 180 г/л, а количество углевода составляет примерно от 120 до 360 г/л.
Репрезентативными микробными полипептидными источниками углерода являются один или более полипептидов дрожжей или бактерий. Репрезентативными растительными полипептидными источниками углерода являются один или более полипептидов сои, кукурузы, канолы, ятрофы, пальмы, арахиса, подсолнечника, кокосового ореха, горчицы, рапса, семян хлопчатника, косточек плодов пальмы, плодов маслины, сафлора, кунжута или семян льна.
Репрезентативными возобновляемыми источниками углерода являются пермеат молочной сыворотки сыра, сироп, получаемый из выжимок кукурузы, меласса сахарной свеклы, солод ячменя и любые их компоненты. Репрезентативными возобновляемыми источниками углерода также являются глюкоза, гексоза, пентоза и ксилоза, присутствующие в биомассе, такой как кукуруза, просо, сахарный тростник, клеточные отходы, образующиеся в процессе ферментации и белковый побочный продукт, образующийся после помола сои, кукурузы или пшеницы. В некоторых вариантах изобретения, источником углерода типа биомассы является лигноцеллюлозный, гемицеллюлозный или целлюлозный материал, такой как, но не ограничивающийся ими, трава, пшеница, пшеничная солома, багасса, багасса сахарного тростника, мягкая древесная пульпа, кукуруза, кукурузные початки или кукурузная шелуха, кукурузные зерна, волокно, получаемое из кукурузных зерен, кукурузная солома, просо, продукт, получаемый из рисовой шелухи, или побочный продукт сырого или сухого помола зерен (например, зерна кукурузы, сорго, ржи, тритикале, ячменя, пшеницы и/или барда). Репрезентативными целлюлозными материалами являются древесина, отходы целлюлозно-бумажной промышленности, травянистые растения и плодовая пульпа. В некоторых вариантах изобретения, источником углерода является любая часть растения, такая как стебли, зерно, корни или клубни. В некоторых вариантах изобретения, в качестве источника углерода используются все нижеследующие растения или их части, такие как кукуруза, пшеница, рожь, сорго, тритикале, рис, просо, ячмень, кассава, бобовые культуры, такие как фасоль и горох, картофель, сладкий картофель, бананы, сахарный тростник и/или тапиока. В некоторых вариантах изобретения, источником углерода является гидролизат биомассы, такой как гидролизат биомассы, включающий ксилозу и глюкозу или сахарозу и глюкозу.
В некоторых вариантах изобретения, возобновляемый источник углерода (такой как биомасса), перед его добавлением в среду для культивирования клеток, подвергают предварительной обработке. В некоторых вариантах изобретения, предварительная обработка включает предварительную ферментативную обработку, химическую предварительную обработку или комбинацию ферментативной и химической предварительной обработки (см., например, публикацию Farzaneh et al., Bioresource Technology 96 (18): 2014-2018, 2005; патент США No. 6176176; и патент США No. 6106888; которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание предварительной обработки возобновляемых источников углерода). В некоторых вариантах изобретения, возобновляемый источник углерода, перед его добавлением в среду для культивирования клеток, подвергают частичному или полному гидролизу.
В некоторых вариантах изобретения, возобновляемый источник углерода (такой как кукурузная солома), перед его добавлением в среду для культивирования клеток, подвергают предварительной обработке путем вытягивания аммиачных волокон (AFEX) (см., например, Farzaneh et al., Bioresource Technology 96 (18): 2014-2018, 2005). В процессе предварительной обработки AFEX, возобновляемый источник углерода обрабатывают жидким безводным аммиаком при умеренной температуре (такой как примерно 60°С-100°С) и при высоком давлении (таком как примерно 250-300 фунт/кв. дюйм = 17240-20700 кПа) примерно в течение 5 минут. Затем давление быстро снимают. В этом процессе, объединенные химические и физические эффекты солюбилизации лигнина, гидролиза гемицеллюлозы, декристаллизации целлюлозы и увеличения площади поверхности приводят почти к полному ферментативному превращению целлюлозы и гемицеллюлозы в ферментируемые сахара. Предварительная AFEX-обработка имеет то преимущество, что почти весь аммиак может быть выделен и использован повторно, а оставшаяся его часть служит в качестве источника азота для микроорганизмов в последующих процессах. Кроме того, для предварительной AFEX-обработки не требуется промывка струей воды. Таким образом, выход сухого вещества после AFEX-обработки составляет почти 100%. По своей природе, AFEX представляет собой процесс высушивания досуха. Такой обработанный возобновляемый источник углерода остается стабильным в течение длительного периода времени и может подаваться при очень высокой загрузке твердых веществ в процессах ферментативного гидролиза или ферментации. Целлюлоза и гемицеллюлоза хорошо сохраняются в AFEX-процессах, и не подвергаются или почти не подвергаются разложению. Перед ферментативным гидролизом возобновляемого источника углерода, подвергаемого предварительной AFEX-обработке, не требуется нейтрализации. Ферментативный гидролиз AFEX-обработанных источников углерода приводит к продуцированию осветленных потоков сахара, которые используются для последующей ферментации.
В некоторых вариантах изобретения, концентрация источника углерода (например, возобновляемого источника углерода) эквивалентна по меньшей мере или примерно 0,1, 0,5, 1, 1,5, 2, 3, 4, 5, 10, 15, 20, 30, 40 или 50% глюкозы (масс./об.). Эквивалентное количество глюкозы может быть определено стандартными ВЭЖХ-методами с использованием глюкозы в качестве стандарта для оценки количества глюкозы, генерируемой из источника углерода. В некоторых вариантах изобретения, концентрация источника углерода (например, возобновляемого источника углерода) эквивалентна примерно 0,1-20% глюкозы, например, примерно 0,1-10% глюкозы, примерно 0,5-10% глюкозы, примерно 1-10% глюкозы, примерно 1-5% глюкозы или примерно 1-2% глюкозы.
В некоторых вариантах изобретения, источник углерода включает дрожжевой экстракт или один или более компонентов дрожжевого экстракта. В некоторых вариантах изобретения, концентрация дрожжевого экстракта составляет по меньшей мере 1 грамм дрожжевого экстракта на литр бульона (г/л, где указанный объем бульона включает объем клеточной среды и объем клеток), например, по меньшей мере или примерно 5, 10, 15, 20, 30, 40, 50, 60, 80, 100, 150, 200, 300 г/л или более. В некоторых вариантах изобретения, концентрация дрожжевого экстракта составляет примерно от 1 до 300 г/л, например, примерно от 1 до 200 г/л, примерно от 5 до 200 г/л, примерно от 5 до 100 г/л или примерно от 5 до 60 г/л. В некоторых вариантах изобретения, концентрация включает общее количество дрожжевого экстракта, добавляемого до и/или во время культивирования клеток-хозяев. В некоторых вариантах изобретения, источник углерода включает дрожжевой экстракт (или один или более его компонентов) и другой источник углерода, такой как глюкоза. В некоторых вариантах изобретения, отношение дрожжевого экстракта к другому источнику углерода составляет примерно 1:5, примерно 1:10 или примерно 1:20 (масс/масс).
Кроме того, источником углерода может быть также субстрат с одним атомом углерода, такой как диоксид углерода или метанол. Продуцирование глицерола из источников, имеющих один атом углерода (например, метанола, формальдегида или формиата) было описано для метилотрофных дрожжей (см. публикацию Yamada et al., Agric. Biol. Chem., 53(2) 541-543, 1989, которая во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание источников углерода) и для бактерий (см. публикацию Hunter et. al, Biochemistry, 24, 4148-4155, 1985, которая во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание источников углерода). Эти микроорганизмы могут усваивать соединения с одним атомом углерода в процессе прохождения окислительного пути от метана до формиата, и продуцировать глицерол. Усвоение углерода может осуществляться по пути через монофосфат рибулозы, через серин или через монофосфат ксилулозы (см. публикацию Gottschalk, Bacterial Metabolism, Second Edition, Springer-Verlag: New York, 1986, которая во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание источников углерода). Рибулозо-монофосфатный путь включает конденсацию формиата с рибулозо-5-фосфатом с образованием сахара с 6 атомами углерода, который превращается во фруктозу, и в конечном счете, в продукт с 3 атомами углерода, а именно глицеральдегид-3-фосфат. Аналогичным образом, сериновый путь способствует усвоению соединения с 1 атомом углерода по гликолитическому пути через метилентетрагидрофолат.
Также известно, что метилотрофные организмы, помимо субстратов с одним и двумя атомами углерода, утилизуют ряд других углерод-содержащих соединений, таких как метиламин, глюкозамин и ряд аминокислот, обладающих метаболической активностью. Так, например, известно, что метилотрофные дрожжи утилизуют углерод метиламина с образованием трегалозы или глицерола (см. публикацию Bellion et al., Microb. Growth Cl Compd., [Int. Symp.], 7th ed., 415-32. Editors: Murrell et al., Publisher: Intercept, Andover, UK, 1993, которая во всей своей полноте вводится в настоящее описание посредством ссылки, а в описание источников углерода). Аналогичным образом, в различных видах Candida происходит метаболизм аланина или олеиновой кислоты (см. публикацию Suiter et al., Arch. Microbiol. 153(5), 485-9, 1990, которая во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание источников углерода).
В некоторых вариантах изобретения, клетки культивируют в стандартной среде, содержащей физиологические соли и питательные вещества (см., например, публикации Pourquie, J. et al., Biochemistry and Genetics of Cellulose Degradation, eds. Aubert et al., Academic Press, pp. 71-86, 1988; и Ilmen et al., Appl. Environ. Microbiol. 63:1298-1306, 1997, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание, относящееся к клеточным средам). Репрезентативными культуральными средами являются стандартные коммерчески доступные среды, такие как бульон Луриа-Бертани (LB), декстрозный бульон Сабуро (SD) или бульон с дрожжевой средой (YM). Могут быть также использованы и другие культуральные среды определенного состава или синтетические среды, и соответствующие среды, подходящие для культивирования конкретных клеток-хозяев и известные специалистам в области микробиологии или ферментации.
Клеточная среда, помимо соответствующего источника углерода, должна содержать, если это необходимо, подходящие микроэлементы, соли, кофакторы, буферы и другие компоненты, известные специалистам в данной области и подходящие для выращивания культур или повышения уровня продуцирования изопрена (см., например, заявку WO 2004/033646 и цитируемые там работы, а также заявку WO 96/35796 и цитируемые там работы, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, их в описание, относящееся к клеточным средам и к условиям культивирования клеток). В некоторых вариантах изобретения, если нуклеиновая кислота изопренсинтазы, DXS, IDI и/или пути MVA находится под контролем индуцибельного промотора, то к данной среде желательно добавить индуцирующий агент (например, сахар, соль металла или антимикробное средство) в концентрации, эффективной для индуцирования экспрессии полипептида изопренсинтазы, DXS, IDI и/или пути MVA. В некоторых вариантах изобретения, клеточная среда содержит антибиотик (такой как канамицин), который соответствует нуклеиновой кислоте-маркеру резистентности к антибиотику (такой как нуклеиновая кислота резистентности к канамицину), присутствующей на векторе, содержащем одну или более нуклеиновых кислот DXS, IDI или пути MVA.
Репрезентативные условия культивирования клеток
Материалы и методы, подходящие для поддержания и роста бактериальных культур, хорошо известны специалистам. Описание репрезентативных методов можно найти в публикациях Manual of Methods for General Bacteriology Gerhardt et al., eds), American Society for Microbiology, Washington, D. C. (1994) или Brock in Biotechnology: A Textbook of Industrial Microbiology, Second Edition (1989) Sinauer Associates, Inc., Sunderland, MA, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание методов культивирования клеток. В некоторых вариантах изобретения, клетки культивируют в культуральной среде в условиях, позволяющих осуществлять экспрессию одного или более полипептидов изопренсинтазы, DXS, IDI или пути MVA, кодируемых нуклеиновой кислотой, встроенной в клетки-хозяева.
Культивирование клеток может быть осуществлено в стандартных условиях культивирования (см., например, заявку WO 2004/033646 и цитируемые там работы, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание условий культивирования клеток и ферментации). Клетки культивируют и поддерживают при соответствующей температуре, в соответствующей газовой смеси и при соответствующем pH (например, примерно при 20-37°C, примерно в 6%-84% CO2, и при pH приблизительно 5-9). В некоторых вариантах изобретения, клетки культивируют при 35°C в соответствующей клеточной среде. В некоторых вариантах изобретения, например, клетки культивируют примерно при 28°C в соответствующей среде в шейкерных культурах или в ферментерах до достижения нужного количества продуцируемого изопрена. В некоторых вариантах изобретения, pH для ферментации составляет в пределах примерно от pH 5,0 до pH 9,0 (например, примерно от 6,0 до 8,0 или примерно от 6,5 до 7,0). Реакции могут быть осуществлены в аэробных, безкислородных или в анаэробных условиях в зависимости от потребностей клеток-хозяев. Репрезентативные условия культивирования данных нитчатых грибов известны специалистам и их описание можно найти в научной литературе и/или в документированных источниках информации о грибах, таких как Американская коллекция типовых культур и Генетический фондовый центр по исследованию грибов (Fungal Genetics Stock Center).
В различных вариантах изобретения, клетки культивируют любым известным способом ферментации, таким как ферментация в периодической культуре, ферментация в периодической культуре с подпиткой или ферментация в непрерывной культуре. В некоторых вариантах изобретения, применяется метод ферментации в периодической культуре. Классическую ферментацию в периодической культуре осуществляют в закрытой системе, где состав среды определяют в начале ферментации и не подвергают искусственным изменениям в процессе ферментации. Так, например, в начале ферментации, клеточную среду инокулируют нужными клетками-хозяевами и оставляют для ферментации, ничего не добавляя в систему. Однако, обычно, под ферментацией «в периодической культуре» подразумевают ферментацию в периодической культуре с добавлением источника углерода, и при такой ферментации часто делают попытки регулировать такие параметры, как значение рН и концентрация кислорода. В системах ферментации в периодической культуре, состав метаболитов и биомассы системы постоянно изменяется вплоть до завершения ферментации. В периодических культурах, клетки проходят через статическую lag-фазу вплоть до логарифмической фазы усиленного роста и наконец до стационарной фазы, при которой рост уменьшается или прекращается. В некоторых вариантах изобретения, клетки в логарифмической фазе ответственны за объем продуцирования изопрена. В некоторых вариантах изобретения, клетки в стационарной фазе продуцируют изопрен.
В некоторых вариантах изобретения используется модифицированная стандартная система ферментации в периодической культуре, такая как система ферментации в периодической культуре с подпиткой. Процессы ферментации в периодической культуре с подпиткой осуществляют в типичной периодической системе, за исключением того, что источник углерода добавляют по мере усиления прохождения процесса ферментации. Системы ферментации в периодической культуре с подпиткой могут быть подходящими в том случае, когда подавление катаболита может ингибировать метаболизм клеток и в том случае, когда желательно использовать ограниченное количество источника углерода в клеточной среде. Ферментация в периодической культуре с подпиткой может быть осуществлена с использованием источника углерода (например, глюкозы) в ограниченном или избыточном количестве. Измерение фактической концентрации источника углерода в системах периодической культуры с подпиткой достаточно затруднено, а поэтому его проводят на основе изменений измеряемых параметров, таких как рН, растворенный кислород и парциальное давление отработанных газов, таких как CO2. Ферментация в периодической культуре и в периодической культуре с подпиткой являются стандартными процедурами, хорошо известными специалистам, и примеры такой ферментации можно найти в публикации: Brock, Biotechnology: A Textbook of Industrial Microbiology, Second Edition (1989) Sinauer Associates, Inc., которая во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание условий культивирования клеток и ферментации.
В некоторых вариантах изобретения используются методы непрерывной ферментации. Непрерывная ферментация представляет собой открытую систему, при которой в биореактор непрерывно подается определенная сбраживаемая среда, и одновременно удаляется равное количество кондиционированной среды, которая затем поступает на ферментативную обработку. При непрерывной ферментации, культуры обычно поддерживаются при постоянной высокой плотности, где клетки находятся, главным образом, в логарифмической фазе роста.
Ферментация в непрерывной культуре позволяет модулировать один параметр или любой из ряда параметров, которые влияют на рост клеток или продуцирование изопрена. Так, например, один из методов позволяет поддерживать ограниченное количество питательных веществ, таких как источник углерода или азот на фиксированном уровне, при сохранении других параметров. В других системах, ряд параметров, влияющих на рост клеток, может постоянно изменяться, а концентрация клеток (например, концентрация, измеренная по мутности среды) остается постоянной. При этом, непрерывные системы желательно поддерживать в стационарной фазе роста. Таким образом, потеря клеток, вызываемая удалением среды, компенсируется скоростью роста клеток при ферментации. Методы модуляции питательных веществ и факторов роста для ферментации в непрерывной культуре, а также методы максимизации скорости образования продукта хорошо известны специалистам в области промышленной микробиологии, и ряд таких методов подробно описан в публикации Brock, Biotechnology: A Textbook of Industrial Microbiology, Second Edition (1989) Sinauer Associates, Inc., которая во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание условий культивирования клеток и ферментации.
В некоторых вариантах изобретения, клетки иммобилизуют на субстрате в качестве целых клеток-катализаторов и создают условия ферментации, подходящие для продуцирования изопрена.
В некоторых вариантах изобретения, бутыли с жидкой культурой помещают в шейкеры в целях введения кислорода в жидкость и сохранения однородности культуры. В некоторых вариантах изобретения, для регуляции температуры, влажности, скорости встряхивания и/или других условий выращивания культуры используют инкубатор. Самыми простыми инкубаторами являются изолированные камеры с регулятором температуры, обычно доводимой до -65°С. Более усовершенствованные инкубаторы также позволяют снижать температуру (путем охлаждения) или регулировать влажность или уровни CO2. Большинство инкубаторов включают таймер, а некоторые из них могут быть также запрограммированы на прохождение циклов при различных температурах, уровнях влажности и т.п. Инкубаторы могут иметь различные размеры, то есть они могут быть настольными или представлять собой установку размером с небольшую комнату.
При желании, часть клеточной среды или вся клеточная среда может быть заменена для пополнения питательных элементов и/или для предотвращения образования потенциально опасных побочных продуктов метаболизма и гибели клеток. В случае суспензионных культур, клетки могут быть отделены от среды путем центрифугирования или фильтрации суспензионной культуры, а затем ресуспендирования клеток в свежей среде. В случае адгезивных культур, среда может быть удалена непосредственно путем отсасывания и замены. В некоторых вариантах изобретения, клеточная среда способствует делению по меньшей мере части клеток, например, осуществлению по меньшей мере или примерно 5, 10, 20, 40, 50, 60, 65 или более клеточных делений в непрерывной культуре (такой как непрерывная культура без разведения).
В некоторых вариантах изобретения, для индуцирования экспрессии нуклеиновой(ых) кислоты (кислот) изопренсинтазы, DXS, IDI или пути MVA, функционально присоединенной(ых) к промотору, используют конститутивный или «ликовый» промотор (такой как промотор Trc), а соединение (такое как IPTG) не добавляют. В некоторых вариантах изобретения, для индуцирования экспрессии нуклеиновой(ых) кислоты (кислот) изопренсинтазы, DXS, IDI или пути MVA, функционально присоединенной(ых) к промотору, добавляют соединение (такое как IPTG).
Репрезентативные способы предотвращения снижения уровня продуцирования изопрена, связанного с ростом клеток
Желательно, чтобы углерод, присутствующий в кормовой биомассе, превращался в изопрен, а не способствовал росту и поддержанию клеток. В некоторых вариантах изобретения, клетки культивируют до низкой или средней OD600, после чего начинается или усиливается продуцирование изопрена. Такая стратегия обеспечивает превращение значительной части углерода в изопрен.
В некоторых вариантах изобретения, клетки достигают определенной оптической плотности, при которой они больше не делятся или делятся крайне медленно, но при этом, продолжают продуцировать изопрен в течение нескольких часов (например, в течение примерно 2, 4, 6, 8, 10, 15, 20, 25, 30 или более часов). Так, например, на фиг. 60A-67C показано, что клетки могут продолжать продуцировать значительное количество мевалоновой кислоты или изопрена после достижения ими оптической плотности, при которой они прекращают свое деление или делятся крайне медленно. В некоторых случаях, оптическая плотность на 550 нм со временем снижается (например, оптическая плотность снижается после того как клетки больше не находятся в экспоненциальной фазе роста, что обусловлено лизисом клеток, прекращением их роста, отсутствием питательных элементов или другими факторами, приводящими к прекращению роста клеток), и эти клетки продолжают продуцировать значительное количество мевалоновой кислоты или изопрена. В некоторых вариантах изобретения, оптическая плотность клеток на 550 нм увеличивается менее чем или примерно на 50% (например, менее чем или примерно на 40, 30, 20, 10, 5 или 0%) в течение определенного периода времени (например, в течение более чем или примерно 5, 10, 15, 20, 25, 30, 40, 50 или 60 часов), и эти клетки продуцируют изопрен в количестве более чем или примерно 1, 10, 25, 50, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900, 1000; 1250; 1500; 1750; 2000; 2500; 3000; 4000; 5000; 10000; 20000; 30000; 40000; 50000; 100000; 200000; 300000; 400000; 500000; 600000; 700000; 800000; 900000; 1000000 или более нмоль изопрена/грамм клеток на сырую массу клеток/час (нмоль/гwcm/час) в течение данного периода времени. В некоторых вариантах изобретения, количество изопрена составляет примерно от 2 до 5000 нмоль/гwcm/час, например, примерно от 2 до 100 нмоль/гwcm/час, примерно от 100 до 500 нмоль/гwcm/час, примерно от 150 до 500 нмоль/гwcm/час, примерно от 500 до 1000 нмоль/гwcm/час, примерно от 1000 до 2000 нмоль/гwcm/час, или примерно от 2000 до 5000 нмоль/гwcm/час. В некоторых вариантах изобретения, количество изопрена составляет примерно от 20 до 5000 нмоль/гwcm/час, примерно от 100 до 5000 нмоль/гwcm/час, примерно от 200 до 2000 нмоль/гwcm/час, примерно от 200 до 1000 нмоль/гwcm/час, примерно от 300 до 1000 нмоль/гwcm/час или примерно от 400 до 1000 нмоль/гwcm/час.
В некоторых вариантах изобретения, оптическая плотность клеток на 550 нм увеличивается на менее чем или примерно на 50% (например, на менее чем или примерно на 40, 30, 20, 10, 5, или 0%) в течение определенного периода времени (например, более чем или примерно в течение 5, 10, 15, 20, 25, 30, 40, 50 или 60 часов), и эти клетки продуцируют кумулятивный титр (общее количество) изопрена в количестве более чем или примерно 1, 10, 25, 50, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900, 1000, 1250, 1500, 1750, 2000, 2500, 3000, 4000, 5000, 10000, 50000, 100000 или более мг изопрена/л бульона (мг/лбульона, где объем бульона включает объем клеток и клеточной среды) в течение этого периода времени. В некоторых вариантах изобретения, количество изопрена составляет примерно от 2 до 5000 мг/лбульона, например, примерно от 2 до 100 мг/лбульона, примерно от 100 до 500 мг/лбульона, примерно от 500 до 1000 мг/лбульона, примерно от 1000 до 2000 мг/лбульона, или примерно от 2000 до 5000 мг/лбульона. В некоторых вариантах изобретения, количество изопрена составляет примерно от 20 до 5000 мг/лбульона, примерно от 100 до 5000 мг/лбульона, примерно от 200 до 2000 мг/лбульона, примерно от 200 до 1000 мг/лбульона, примерно от 300 до 1000 мг/лбульона, или примерно от 400 до 1000 мг/лбульона.
В некоторых вариантах изобретения, оптическая плотность клеток на 550 нм увеличивается на менее чем или примерно на 50% (например, на менее чем или примерно на 40, 30, 20, 10, 5, или 0%) в течение определенного периода времени (например, более чем или примерно в течение 5, 10, 15, 20, 25, 30, 40, 50 или 60 часов), и эти клетки превращают более чем или примерно 0,0015, 0,002, 0,005, 0,01, 0,02, 0,05, 0,1, 0,12, 0,14, 0,16, 0,2, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9, 1,0, 1,2, 1,4, 1,6, 1,8, 2,0, 2,5, 3,0, 3,5, 4,0, 5,0, 6,0, 7,0 или 8,0% углерода в культуральной клеточной среде в изопрен в течение данного периода времени. В некоторых вариантах изобретения, процент превращения углерода в изопрен составляет примерно от 0,002 до 4,0%, примерно от 0,002 до 3,0%, примерно от 0,002 до 2,0%, примерно от 0,002 до 1,6%, примерно от 0,002 до 0,005%, примерно от 0,005 до 0,01%, примерно от 0,01 до 0,05%, примерно от 0,05 до 0,15%, от 0,15 до примерно 0,2%, примерно от 0,2 до 0,3%, примерно от 0,3 до 0,5%, примерно от 0,5 до 0,8%, примерно от 0,8 до 1,0%, или примерно от 1,0 до 1,6%. В некоторых вариантах изобретения, процент превращения углерода в изопрен составляет примерно от 0,002 до 0,4%, от 0,002 до примерно 0,16%, от 0,04 до примерно 0,16%, примерно от 0,005 до 0,3%, примерно от 0,01 до 0,3%, или примерно от 0,05 до 0,3%.
В некоторых вариантах изобретения, изопрен продуцируется только в стационарной фазе. В некоторых вариантах изобретения, изопрен продуцируется в фазе роста и в стационарной фазе. В различных вариантах изобретения, количество продуцируемого изопрена (например, общее количество продуцируемого изопрена или количество продуцируемого изопрена на литр бульона в час на OD600) в стационарной фазе по меньшей мере или примерно в 2, 3, 4, 5, 10, 20, 30, 40, 50 или более раз превышает количество продуцируемого изопрена в фазе роста в течение того же самого периода времени. В различных вариантах изобретения, более чем или примерно 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 95, 99% или более от общего количества продуцируемого изопрена (например, изопрена, продуцируемого в процессе ферментации в течение определенного периода времени, например, 20 часов) продуцируется в клетках, находящихся в стационарной фазе роста. В различных вариантах изобретения, более чем или примерно 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 95, 99% или более от общего количества продуцируемого изопрена (например, изопрена, продуцируемого в процессе ферментации в течение определенного периода времени, например, 20 часов) продуцируется в клетках, которые медленно делятся или вообще не делятся, например, в клетках, у которых оптическая плотность на 550 нм увеличивается менее чем или примерно на 50% (например, менее чем или примерно на 40, 30, 20, 10, 5 или 0%). В некоторых вариантах изобретения, изопрен продуцируется только в фазе роста.
В некоторых вариантах изобретения, одну или более нуклеиновых кислот пути MVA, а также нуклеиновых кислот IDI, DXP или изопренсинтазы помещают под контроль промотора или фактора, который является более активным в стационарной фазе, чем в фазе роста. Так, например, одна или более нуклеиновых кислот пути MVA, а также нуклеиновых кислот IDI, DXP или изопренсинтазы могут находиться под контролем фактора сигма в стационарной фазе, такого как RpoS. В некоторых вариантах изобретения, одна или более нуклеиновых кислот пути MVA, а также нуклеиновых кислот IDI, DXP или изопренсинтазы находятся под контролем промотора, который является индуцибельным в стационарной фазе, такого как промотор, индуцируемый регулятором ответа, который является активным в стационарной фазе.
Продуцирование изопрена в интервалах безопасных рабочих уровней
Принимая во внимание воспламеняемость изопрена, очевидно, что его продуцирование на безопасных рабочих уровнях значительно облегчит разработку и конструирование промышленного оборудования, повысит степень безопасности работы с ним и ограничит вероятность возгорания изопрена. В частности, оптимальные интервалы продуцирования изопрена определяют зону безопасности, то есть те интервалы концентрации изопрена, которые не вызывают воспламенения. В одном из таких аспектов, настоящее изобретение относится к способу продуцирования изопрена в интервале концентраций, не вызывающих воспламенения (за пределами огибающей кривой воспламеняемости изопрена).
Так, например, для определения пределов воспламеняемости изопрена (например, изопрена в присутствии O2, N2, CO2, или любой комбинации двух или более вышеуказанных газов) в целях гарантии безопасности проводят компьютерное моделирование и экспериментальные испытания. Огибающая кривая воспламеняемости характеризуется низким пределом воспламеняемости (LFL), верхним пределом воспламеняемости (UFL), ограничением концентрации кислорода (LOC) и ограничением температуры. Для воспламенения системы, минимальное количество топлива (такого как изопрен) должно находиться в присутствии минимального количества окислителя, обычно кислорода. LFL представляет собой минимальное количество изопрена, которое должно присутствовать для поддержания горения, а UFL представляет собой максимальное количество изопрена, которое может присутствовать. Выше этого предела, смесь представляет собой обогащенное топливо, и при этом, фракция кислорода должна быть слишком мала для образования воспламеняющей смеси. LOC означает минимальную фракцию кислорода, которая также должна присутствовать для образования воспламеняющей смеси. Ограничение температуры должно исходить из точки вспышки изопрена и наименьшей температуры, при которой может распространяться пламя, образующееся при горении изопрена. Эти пределы зависят от концентрации изопрена, типа и концентрации окислителя, инертных газов, присутствующих в системе, температуры и давлении системы. Составы, которые входят в пределы огибающей кривой воспламеняемости, способны поддерживать горение и требуют дополнительных мер предосторожности при конструировании и эксплуатации оборудования, работающего с использованием этих составов.
Нижеследующие условия были протестированы с применением компьютерного моделирования, математического анализа и экспериментального тестирования. При желании, для определения LFL, UFL и концентраций LOC могут быть протестированы и другие условия (такие как другие температуры, давление и составы постоянного газа) с применением методов, описанных здесь для определения концентраций.
(1) Компьютерное моделирование и математический анализ
Серия испытаний 1: изопрен: 0 масс.% - 14 масс.%;
O2: 6 масс.% - 21 масс.%;
N2: 79 масс.% - 94 масс.%;
Серия испытаний 2: изопрен: 0 масс.% - 14 масс.%;
O2: 6 масс.% - 21 масс.%;
N2: 79 масс.% - 94 масс.%;
насыщение H2O
Серия испытаний 3: изопрен: 0 масс.% - 14 масс.%;
O2: 6 масс.% - 21 масс.%;
N2: 79 масс.% - 94 масс.%;
CO2: 5 масс.% - 30 масс.%
(2) Экспериментальные испытания для конечного определения пределов воспламеняемости
Серия испытаний 1: изопрен: 0 масс.% - 14 масс.%;
O2: 6 масс.% - 21 масс.%;
N2: 79 масс.% - 94 масс.%
Серия испытаний 2: изопрен: 0 масс.% - 14 масс.%;
O2: 6 масс.% - 21 масс.%;
N2: 79 масс.% - 94 масс.%;
насыщение H2O
Для оценки воспламеняемости системы в нескольких различных условиях испытания проводили компьютерное моделирование. CO2 не оказывал какого-либо значимого влияния на пределы воспламеняемости системы. Наборы тестов 1 и 2 были созданы путем экспериментального тестирования. Результаты моделирования совпадали с результатами экспериментальных тестов. Было обнаружено, что добавление воды приводит лишь к незначительным изменениям.
Как было определено, LOC составляет 9,5 об.% для смеси изопрена, O2, N2 и CO2 при 40°C и при давлении в 1 атмосферу. Добавление до 30% CO2 не оказывало значительного влияния на воспламеняемость смеси изопрена, O2 и N2. Было показано, что между безводной и насыщенной водной системой изопрена, O2 и N2 наблюдались лишь незначительные изменения воспламеняемости. Нижний предел температур составляет примерно -54°C. Температура ниже примерно -54°C является слишком низкой для поддержания горения изопрена.
В некоторых вариантах изобретения, LFL изопрена составляет в пределах примерно от 1,5 об.% до 2,0 об.%, а UFL изопрена составляет в пределах примерно от 2,0 об.% до 12,0 об.% в зависимости от количества кислорода в системе. В некоторых вариантах изобретения, LOC составляет примерно 9,5 об.% кислорода. В некоторых вариантах изобретения, LFL изопрена составляет примерно от 1,5 об.% до 2,0 об.%, UFL изопрена составляет примерно от 2,0 об.% до 12,0 об.%, а LOC составляет примерно 9,5 об.% кислорода при температуре примерно от 25°C до 55°C (например, примерно 40°C) и давлении примерно от 1 атмосферы до 3 атмосфер.
В некоторых вариантах изобретения, изопрен продуцируется в присутствии менее чем примерно 9,5 об.% кислорода (то есть при LOC ниже величины, необходимой для воспламеняемости смеси изопрена). В некоторых вариантах изобретения, в которых изопрен продуцируется в присутствии более чем или примерно 9,5 об.% кислорода, концентрация изопрена ниже LFL (например, примерно ниже 1,5 об.%). Так, например, количество изопрена может поддерживаться ниже LFL путем разбавления композиции изопрена инертным газом (например, постоянного или периодического добавления инертного газа, такого как азот, для поддержания композиции изопрена ниже LFL). В некоторых вариантах изобретения, в которых изопрен продуцируется в присутствии более чем или примерно 9,5 об.% кислорода, концентрация изопрена выше UFL (например, примерно выше 12 об.%). Так, например, количество изопрена может поддерживаться выше UFL с использованием системы (такой как любая из описанных здесь систем культивирования клеток), которая продуцирует изопрен в концентрации выше UFL. При желании, может быть использован относительно низкий уровень кислорода, то есть такой уровень, при котором UFL также будет относительно низким. В этом случае, для поддержания более высокого значения UFL необходима более низкая концентрация изопрена.
В некоторых вариантах изобретения, в которых изопрен продуцируется в присутствии более чем или примерно 9,5 об.% кислорода, концентрация изопрена входит в пределы огибающей кривой воспламеняемости (например, в пределы между значениями LFL и UFL). В некоторых вариантах изобретения, когда концентрация изопрена может находиться в пределах огибающей кривой воспламеняемости, необходимо принять одну или более мер по снижению вероятности пожара или взрыва. Так, например, может быть устранен один или более источников возгорания (такой как любой материал, который может вызывать искру). В некоторых вариантах изобретения, принимают одну или более мер по снижению времени, в течение которого концентрация изопрена сохраняется в пределах огибающей кривой воспламеняемости. В некоторых вариантах изобретения, для того, чтобы определить, близка ли концентрация изопрена к огибающей кривой воспламеняемости, или входит в нее, используют датчик. При желании, концентрация изопрена может быть измерена в течение одного или более периодов времени в процессе культивирования клеток, а условия культивирования клеток и/или количество инертного газа могут быть скорректированы стандартными методами, в случае, если концентрация изопрена близка к огибающей кривой воспламеняемости, или входит в нее. В конкретных вариантах изобретения, условия культивирования клеток (такие как условия ферментации) корректируют в целях снижения концентрации изопрена ниже LFL или увеличения концентрации изопрена выше UFL. В некоторых вариантах изобретения, количество изопрена поддерживается ниже LFL путем разбавления композиции изопрена инертным газом (например, постоянного или периодического добавления инертного газа, такого как азот, для поддержания композиции изопрена ниже LFL).
В некоторых вариантах изобретения, количество воспламеняющихся летучих веществ, не являющихся изопреном (таких как один или более сахаров), по меньшей мере примерно в 2, 5, 10, 50, 75 или 100 раз ниже количества продуцируемого изопрена. В некоторых вариантах изобретения, часть газовой фазы, не являющейся изопреном, содержит примерно от 0% до 100% (по объему) кислорода, например, примерно от 0% до 10%, примерно от 10% до 20%, примерно от 20% до 30%, примерно от 30% до 40%, примерно от 40% до 50%, примерно от 50% до 60%, примерно от 60% до 70%, примерно от 70% до 80%, примерно от 80% до 90%, или примерно от 90% до 100% (по объему) кислорода. В некоторых вариантах изобретения, часть газовой фазы, не являющейся изопреном, содержит примерно от 0% до 99% (по объему) азота, например, примерно от 0% до 10%, примерно от 10% до 20%, примерно от 20% до 30%, примерно от 30% до 40%, примерно от 40% до 50%, примерно от 50% до 60%, примерно от 60% до 70%, примерно от 70% до 80%, примерно от 80% до 90%, или примерно от 90% до 99% (по объему) азота.
В некоторых вариантах изобретения, часть газовой фазы, не являющейся изопреном, содержит примерно от 1% до 50% (по объему) CO2, например, примерно от 1% до 10%, примерно от 10% до 20%, примерно от 20% до 30%, примерно от 30% до 40% или примерно от 40% до 50% (по объему) CO2.
В некоторых вариантах изобретения, композиция изопрена также содержит этанол. Так, например, этанол может быть использован для экстрактивной перегонки изопрена, что приводит к получению композиций (таких как потоки промежуточного продукта), которые включают этанол и изопрен. При этом, желательно, чтобы количество этанола входило в пределы огибающей кривой воспламеняемости. LOC этанола составляет примерно 8,7 об.%, а LFL для этанола составляет от 3,3 об.% в стандартных условиях, таких как давление примерно в 1 атмосферу и температура примерно 60°F (см. публикацию NFPA 69 Standard on Explosion Prevention Systems, издание 2008, которое во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание величин LOC, LFL и UFL). В некоторых вариантах изобретения, композиции, включающие изопрен и этанол, получают в присутствии этанола в концентрации, меньшей, чем LOC, которая необходима для того, чтобы смесь этанола становилась воспламеняемой (например, менее чем примерно 8,7% об.%). В некоторых вариантах, в которых композиции, включающие изопрен и этанол, продуцируют в присутствии этанола в концентрации, превышающей или примерно составляющей концентрацию LOC, необходимую для того, чтобы смесь этанола становилась воспламеняемой, то есть концентрация этанола ниже LFL (такой как менее чем примерно 3,3 об.%).
В различных вариантах изобретения, количество окислителя (такого как кислород) ниже LOC любого топлива в данной системе (такого как изопрен или этанол). В различных вариантах изобретения, количество окислителя (такого как кислород) примерно на 60, 40, 30, 20, 10 или 5% ниже LOC изопрена или этанола. В различных вариантах изобретения, количество окислителя (такого как кислород) по меньшей мере на 2, 4, 5 или более абсолютных процентных точек (об.%) ниже LOC изопрена или этанола. В конкретных вариантах изобретения, количество кислорода по меньшей мере на 2 абсолютные процентные точки (об.%) меньше, чем LOC изопрена или этанола (например, концентрация кислорода составляет менее чем 7,5 об.%, если LOC изопрена составляет 9,5 об.%). В различных вариантах изобретения, количество топлива (такого как изопрен или этанол) составляет менее чем или примерно 25, 20, 15, 10 или 5% от величины LFL для этого топлива.
Примеры продуцирования изопрена
В некоторых вариантах изобретения, клетки культивируют в культуральной среде в условиях, стимулирующих продуцирование изопрена этими клетками. Термин «пик абсолютной продуктивности» означает максимальное абсолютное количество изопрена в отходящем газе в процессе культивирования клеток в течение конкретного периода времени (например, культивирования клеток в процессе конкретной процедуры ферментации). Термин «пик абсолютной продуктивности в данный момент времени» означает момент времени в процессе ферментации, когда абсолютное количество изопрена в отходящем газе является максимальным в процессе культивирования клеток в течение конкретного периода времени (например, культивирования клеток в процессе конкретной процедуры ферментации). В некоторых вариантах изобретения, количество изопрена измеряют в момент времени, в который имеет место максимальная абсолютная продуктивность. В некоторых вариантах изобретения, пик абсолютной продуктивности для клеток представляет собой приблизительно любое количество изопрена, описанное в настоящей заявке.
Термин «пик удельной продуктивности» означает максимальное количество продуцируемого изопрена на клетку в процессе культивирования клеток в течение конкретного периода времени (например, культивирования клеток в процессе конкретной процедуры ферментации). Термин «пик удельной продуктивности в данный момент времени» означает момент определенного периода времени в процессе культивирования клеток (например, культивирования клеток в процессе конкретной процедуры ферментации), когда количество продуцируемого изопрена на клетку достигает максимума. Удельную продуктивность определяют путем деления общей продуктивности на количество клеток, измеренное при оптической плотности на 600 нм (OD600). В некоторых вариантах изобретения, количество изопрена определяют при максимальной удельной продуктивности в данный момент времени. В некоторых вариантах изобретения, пик удельной продуктивности для клеток представляет собой приблизительно любое количество изопрена, описанное в настоящей заявке.
Термин «кумулятивная общая продуктивность» означает кумулятивное общее количество продуцируемого изопрена в процессе культивирования клеток в течение конкретного периода времени (например, культивирования клеток в процессе конкретной процедуры ферментации). В некоторых вариантах изобретения, измеряют кумулятивное общее количество изопрена. В некоторых вариантах изобретения, кумулятивная общая продуктивность для клеток представляет собой приблизительно любое количество изопрена, описанное в настоящей заявке.
Термин «относительный детектируемый ответ» означает отношение детектируемого ответа (такого как площадь ГХ/МС) для одного соединения (такого как изопрен) к детектируемому ответу (такому как площадь ГХ/МС) одного или более соединений (таких как все C5-углеводороды). Детектируемый ответ может быть измерен, как описано в настоящей заявке, например, с помощью ГХ/МС-анализа, проводимого с использованием системы ГХ/МС Agilent 6890, снабженной колонкой для ГХ/МС Agilent HP-5MS (30 м × 250 мкм; толщина пленки 0,25 мкм). При желании, относительный детектируемый ответ может быть преобразован в процент по массе с использованием параметров ответа для каждого из соединений. Таким параметром ответа является показатель уровня генерирования сигнала для данного количества конкретного соединения (то есть показатель чувствительности детектора к конкретному соединению). Такой параметр ответа может быть использован в качестве коррелляционного коэффициента для преобразования относительного детектируемой ответа в процент по массе, если детектор имеет другую чувствительность к сравниваемым соединениям. Альтернативно, процент по массе может быть аппроксимирован, если предположить, что параметры ответа являются одинаковыми для сравниваемых соединений. Таким образом, можно предположить, что процент по массе является приблизительно таким же, как и относительный детектируемый ответ.
В некоторых вариантах изобретения, клетки в культуре продуцируют изопрен в количестве, превышающем или составляющем примерно 1, 10, 25, 50, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900, 1000; 1250; 1500; 1750; 2000; 2500; 3000; 4000; 5000 или более нмоль изопрена/грамм клеток на сырую массу клеток/час (нмоль/гwcm/час). В некоторых вариантах изобретения, количество изопрена составляет примерно от 2 до 5000 нмоль/гwcm/час, например, примерно от 2 до 100 нмоль/гwcm/час, примерно от 100 до 500 нмоль/гwcm/час, примерно от 150 до 500 нмоль/гwcm/час, примерно от 500 до 1000 нмоль/гwcm/час, примерно от 1000 до 2000 нмоль/гwcm/час, или примерно от 2000 до 5000 нмоль/гwcm/час. В некоторых вариантах изобретения, количество изопрена составляет примерно от 20 до 5000 нмоль/гwcm/час, примерно от 100 до 5000 нмоль/гwcm/час, примерно от 200 до 2000 нмоль/гwcm/час, примерно от 200 до 1000 нмоль/гwcm/час, примерно от 300 до 1000 нмоль/гwcm/час, или примерно от 400 до 1000 нмоль/гwcm/час.
Количество изопрена, выраженное в нмоль/гwcm/час, может быть измерено, как описано в патенте США No. 5849970, который во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание измерения уровня продуцирования изопрена. Так, например, два миллилитра (например, зоны «хэдспейс» от культуры, такой как 2 мл культуры, выращенной в герметично закрытых сосудах при 32°C со встряхиванием при 200 об/мин в течение приблизительно 3 часов) анализируют на уровни изопрена с использованием стандартной системы газовой хроматографии, такой как система, работающая в изотермических условиях (85°C) и снабженная колонкой с н-октаном/порасилом С (Alltech Associates, Inc., Deerfield, I11.), а также подсоединенная к газовому детектору, в котором в качестве восстановителя используется оксид ртути RGD2 (Trace Analytical, Menlo Park, CA) (см., например, публикации Greenberg et al., Atmos. Environ. 27A: 2689-2692, 1993; Silver et al., Plant Physiol. 97:1588-1591, 1991, которые вводятся в настоящее описание посредством ссылки, а в частности, в описание, относящееся к измерению уровня продуцирования изопрена). Величины площади хроматограмм, полученные в газовой хроматографии, преобразуют в нмоль изопрена с использованием стандартной калибровочной кривой для концентраций изопрена. В некоторых вариантах изобретения, массу клеток в граммах в расчете на сырую массу вычисляют путем определения величины A600 для образца клеточной культуры, а затем эту величину преобразуют в величину A600 в граммах клеток, исходя из калибровочной кривой зависимости сырой массы клеточных культур от известной величины A600. В некоторых вариантах изобретения, оценку количества клеток в граммах проводят при предположении, что один литр бульона (включая клеточную среду и клетки), имеющего величину A600=1, имеет 1 грамм сырой клеточной массы. Эту величину также делят на количество времени (часов) инкубирования культуры, например, трех часов.
В некоторых вариантах изобретения, клетки в культуре продуцируют изопрен в количестве более чем или примерно 1, 10, 25, 50, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900, 1000; 1250; 1500; 1750; 2000; 2500; 3000; 4000; 5000; 10000; 1000000 или более нмоль изопрена/грамм клеток на сырую массу клеток/час (нмоль/гwcm/час). В некоторых вариантах изобретения, количество изопрена составляет примерно от 2 до 5000 нмоль/гwcm/час, например, примерно от 2 до 100 нмоль/гwcm/час, примерно от 100 до 500 нмоль/гwcm/час, примерно от 150 до 500 нмоль/гwcm/час, примерно от 500 до 1000 нмоль/гwcm/час, примерно от 1000 до 2000 нмоль/гwcm/час, или примерно от 2000 до 5000 нмоль/гwcm/час. В некоторых вариантах изобретения, количество изопрена составляет примерно от 20 до 5000 нмоль/гwcm/час, примерно от 100 до 5000 нмоль/гwcm/час, примерно от 200 до 2000 нмоль/гwcm/час, примерно от 200 до 1000 нмоль/гwcm/час, примерно от 300 до 1000 нмоль/гwcm/час, или примерно от 400 до 1000 нмоль/гwcm/час. Количество изопрена, выраженное в нг/гwcm/час, может быть вычислено путем умножения величины для продуцирования изопрена, полученной в нмоль/гwcm/час, на 68,1, как обсуждалось выше (как указано ниже в уравнении 5).
В некоторых вариантах изобретения, клетки в культуре продуцируют кумулятивный титр (общее количество) изопрена в количестве более чем или примерно 1, 10, 25, 50, 100, 150, 200, 250, 300, 400, 500, 600, 700, 800, 900, 1000, 1250, 1500, 1750, 2000, 2500, 3000, 4000, 5000, 10000, 50000, 100000 или более мг изопрена/л бульона (мг/лбульона, где объем бульона включает объем клеток и клеточной среды). В некоторых вариантах изобретения, количество изопрена составляет примерно от 2 до 5000 мг/лбульона, например, примерно от 2 до 100 мг/лбульона, примерно от 100 до 500 мг/лбульона, примерно от 500 до 1000 мг/лбульона, примерно от 1000 до 2000 мг/лбульона, или примерно от 2000 до 5000 мг/лбульона. В некоторых вариантах изобретения, количество изопрена составляет примерно от 20 до 5000 мг/лбульона, примерно от 100 до 5000 мг/лбульона, примерно от 200 до 2000 мг/лбульона, примерно от 200 до 1000 мг/лбульона, примерно от 300 до 1000 мг/лбульона, или примерно от 400 до 1000 мг/лбульона.
Удельная продуцируемость изопрена, выраженная в мг изопрена/л зоны «хэдспейс» культур в шейкерных колбах или аналогичных культур может быть измерена путем взятия 1 мл образца клеточной культуры с величиной OD600 приблизительно 1,0, и помещения его в 20 мл сосуд, с последующим инкубированием в течение 30 минут и измерением количества изопрена в зоне «хэдспейс» (как описано, например, в примере I, части II). Если величина OD600 не равна 1,0, то измеренная величина может быть нормализована по величине OD600=1,0 путем ее деления на величину OD600. Величина, выраженная в мг изопрена/л зоны «хэдспейс», может быть преобразована в мг/лбульона/час/OD600 бульона культуры путем умножения на 38. Величина, выраженная в единицах мг/лбульона/час/OD600, может быть умножена на количество часов и величину OD600 с получением кумулятивного титра, выраженного в мг изопрена/литр бульона.
Мгновенная скорость продуцирования изопрена, выраженная в мг/лбульона/час в ферментере, может быть измерена путем взятия образца отходящего газа из ферментера, анализа этого образца на количество изопрена (выраженное в мг изопрена на лгаза) как описано, например, в примере I, части II, и умножения этой величины на скорость прохождения отходящего газа через каждый литр бульона (например, при 1 vvm (объем воздуха/объем бульона/минуту), например, на 60 лгаза/час). Таким образом, уровень отходящего газа в 1 мг/лгаза соответствует мгновенной скорости продуцирования 60 мг/лбульона/час в потоке воздуха при 1 vvm. При желании, величина, выраженная в мг/лбульона/час, может быть разделена на величину OD600 с получением удельной скорости, выраженной в мг/лбульона/час/OD. Средняя величина мг изопрена/лгаза может быть преобразована в общую продуцируемость продукта (граммы изопрена на литр сбраживаемого бульона, мг/лбульона) путем умножения средней концентрации отходящего газа изопрена на общее количество барботированного отходящего газа на литр сбраживаемого бульона в процессе ферментации. Таким образом, средняя концентрация отходящего газа изопрена в количестве 0,5 мг/лбульона/час в течение 10 часов при 1 vvm соответствует общей концентрации продукта 300 мг изопрена/лбульона.
В некоторых вариантах изобретения, клетки в культуре превращают более чем или примерно 0,0015, 0,002, 0,005, 0,01, 0,02, 0,05, 0,1, 0,12, 0,14, 0,16, 0,2, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9, 1,0, 1,2, 1,4, 1,6, 1,8, 2,0, 2,5, 3,0, 3,5, 4,0, 5,0, 6,0, 7,0 или 8,0% углерода в культуральной клеточной среде в изопрен. В некоторых вариантах изобретения, процент превращения углерода в изопрен составляет примерно от 0,002 до 4,0%, примерно от 0,002 до 3,0%, примерно от 0,002 до 2,0%, примерно от 0,002 до 1,6%, примерно от 0,002 до 0,005%, примерно от 0,005 до 0,01%, примерно от 0,01 до 0,05%, примерно от 0,05 до 0,15%, от 0,15 до примерно 0,2%, примерно от 0,2 до 0,3%, примерно от 0,3 до 0,5%, примерно от 0,5 до 0,8%, примерно от 0,8 до 1,0%, или примерно от 1,0 до 1,6%. В некоторых вариантах изобретения, процент превращения углерода в изопрен составляет примерно от 0,002 до 0,4%, от 0,002 до примерно 0,16%, от 0,04 до примерно 0,16%, примерно от 0,005 до 0,3%, примерно от 0,01 до примерно 0,3%, или примерно от 0,05 до 0,3%.
Процент превращения углерода в изопрен (также называемый «% выхода углерода») может быть измерен путем деления моль углерода в продуцируемом изопрене на моль углерода в источнике углерода (например, моль углерода в периодической культуре и в культуре с подпиткой глюкозой и дрожжевым экстрактом). Это число умножают на 100% и получают величину в процентах (как указано в уравнении 1).
Уравнение 1
% Выхода углерода = (моль углерода в продуцируемом изопрене)/(моль углерода в источнике углерода)×100
Для вычисления данной величины, можно допустить, что дрожжевой экстракт содержит 50 масс.% углерода. Так, например, для 500 литров, как описано в примере 7, части VIII, процент превращения углерода в изопрен может быть вычислен, как указано в уравнении 2.
Уравнение 2
% Выхода углерода = (39,1 г изопрена × 1/68,1 моль/г × 5 C/моль)/[(181221 г глюкозы × 1/180 моль/г × 6 C/моль)+(17780 г дрожжевого экстракта × 0,5 × 1/12 моль/г)] × 100=0,042%
Для двух 500-литровых ферментеров, описанных в настоящей заявке (пример 7, части VII и VIII), процент превращения углерода в изопрен составляет 0,04-0,06%. 0,11-0,16%-ный выход углерода достигался с использованием 14-литровых систем, описанных в настоящей заявке. В примере 11, части V, описано 1,53%-ное превращение углерода в изопрен с применением описанных здесь методов.
Специалист в данной области может легко преобразовать скорость продуцирования изопрена или количество продуцируемого изопрена в любые другие единицы. Репрезентативные уравнения для взаимопревращения таких единиц приводятся ниже.
Единицы скорости (общей и удельной) продуцирования изопрена
Уравнение 3
1 г изопрена/лбульона/час = 14,7 ммоль изопрена/лбульона/час (общая объемная скорость)
Уравнение 4
1 нмоль изопрена/гwcm/час = 1 нмоль изопрена/лбульона/час/OD600 (при этом преобразовании было принято, что один литр бульона, имеющего величину OD600 = 1, содержит 1 грамм сырой клеточной массы)
Уравнение 5
1 нмоль изопрена/гwcm/час = 68,1 нг изопрена/гwcm/час (в расчете на молекулярную массу изопрена)
Уравнение 6
1 нмоль изопрена/лгаза O2/час = 90 нмоль изопрена/лбульона/час (при скорости потока O2=90 л/час на литр культурального бульона)
Уравнение 7
1 мкг изопрена/лгаза изопрена в отходящем газе = 60 мкг изопрена/лбульона/час при скорости потока 60 лгаза на литр культурального бульона (1 vvm)
Единицы для титра (общего и удельного)
Уравнение 8
1 нмоль изопрена/мг клеточного белка = 150 нмоль изопрена/лбульона/OD600 ((при этом преобразовании было принято, что один литр бульона, имеющего величину OD600=1, содержит приблизительно 150 мг общего клеточного белка)(удельная продуктивность).
Уравнение 9
1 г изопрена/лбульона = 14,7 ммоль изопрена/лбульона (общий титр)
При желании, уравнение 10 может быть использовано для преобразования любых величин, которые включают сырую массу клеток, в соответствующие величины, которые включают сухую массу клеток.
Уравнение 10
Сухая масса клеток = (сырая масса клеток)/3,3
При желании, уравнение 11 может быть использовано для взаимопреобразования величин м.д. и мкг/л. В частности, «м.д.» означает миллионные доли, определенные как мкг/г (масс./масс.) или мкл/л (об./об.).
Превращение мкг/л в м.д. (например, мкг аналита на грамм газа) может быть осуществлено путем определения массы на литр отходящего газа (то есть плотности газа). Так, например, литр воздуха при STP имеет плотность приблизительно 1,2 г/л. Таким образом, концентрация 1 м.д. (мкг/г) равна 0,83 мкг/л при STP (уравнение 11). Преобразование м.д. (мкг/г) в мкг/л зависит от давления, температуры и общего состава отходящего газа.
Уравнение 11
1 м.д. (мкг/г) равно 0,83 мкг/л при стандартной температуре и при стандартном давлении (STP; 101,3 кПа (1 бар) и 273,15K).
Превращение мкг/л в м.д. по объему (например, мкл аналита на литр газа) может быть осуществлено по универсальному газовому закону (уравнение 12). Так, например, концентрация отходящего газа 1000 мкг/лгаза соответствует 14,7 мкмоль/лгаза. Универсальная газовая постоянная составляет 0,082057 л · атм · K-1 ·моль-1, то есть в соответствии с уравнением 12, объем, занимаемый 14,7 мкмоль HG при STP равен 0,329 мл. Поэтому, концентрация 1000 мкг/л HG равна 329 м.д. по объему или 0,0329% (об/об) при STP.
Уравнение 12
PV=nRT, где «P» означает давление, «V» означает объем, «n» означает моли газа, «R» означает универсальную газовую постоянную, а «T» означает температуру по Кельвину.
Количество примесей в изопреновых композициях обычно измеряют в величинах по массе на объем (масс./об.), например, в мкг/л. При желании, единицы измерения, выраженные в мкг/л, могут быть преобразованы в единицы мг/м3 с помощью уравнения 13.
Уравнение 13
1 мкг/л = 1 мг/м3
В некоторых вариантах изобретения, клетки, содержащие гетерологичную нуклеиновую кислоту, кодирующую полипептид изопренсинтазы, продуцируют изопрен в количестве, которое по меньшей мере или примерно в 2, 3, 5, 10, 25, 50, 100, 150, 200, 400 раз или более превышает количество изопрена, продуцируемое из соответствующих клеток, культивированных, в основном, в тех же условиях, но в отсутствии гетерологичной нуклеиновой кислоты, кодирующей полипептид изопренсинтазы.
В некоторых вариантах изобретения, клетки, содержащие гетерологичную нуклеиновую кислоту, кодирующую полипептид изопренсинтазы, и одну или более гетерологичных нуклеиновых кислот, кодирующих полипептид DXS, IDI и/или пути MVA, продуцируют изопрен в количестве, которое по меньшей мере или примерно в 2, 3, 5, 10, 25, 50, 100, 150, 200, 400 раз или более превышает количество изопрена, продуцируемое из соответствующих клеток, культивированных, в основном, в тех же условиях, но в отсутствии гетерологичных нуклеиновых кислот.
В некоторых вариантах изобретения, указанная композиция изопрена включает более чем или примерно 99,90; 99,92; 99,94; 99,96; 99,98 или 100% изопрена по общей массе всех C5-углеводородов в данной композиции. В некоторых вариантах изобретения, указанная композиция дает относительный детектируемый ответ для изопрена, который превышает или составляет примерно 99,90, 99,91, 99,92, 99,93, 99,94, 99,95, 99,96, 99,97, 99,98, 99,99 или 100% по отношению к детектируемому ответу для всех C5-углеводородов в данной композиции. В некоторых вариантах изобретения, композиция изопрена содержит примерно от 99,90 до 99,92, примерно от 99,92 до 99,94, примерно от 99,94 до 99,96, примерно от 99,96 до 99,98, примерно от 99,98 до 100% изопрена по массе всех C5-углеводородов в данной композиции.
В некоторых вариантах изобретения, указанная композиция изопрена включает менее чем или примерно 0,12, 0,10, 0,08, 0,06, 0,04, 0,02, 0,01, 0,005, 0,001, 0,0005, 0,0001, 0,00005 или 0,00001% C5-углеводородов, не являющихся изопреном (таких как 1,3-циклопентадиен, цис-1,3-пентадиен, транс-1,3-пентадиен, 1-пентин, 2-пентин, 1-пентен, 2-метил-1-бутен, 3-метил-1-бутин, транс-пиперилен, цис-пиперилен, пент-4-ен-1-ин, транс-пент-3-ен-1-ин или цис-пент-3-ен-1-ин), по общей массе всех C5-углеводородов в указанной композиции. В некоторых вариантах изобретения, указанная композиция дает относительный детектируемый ответ для C5-углеводородов, не являющихся изопреном, который составляет менее чем или примерно 0,12, 0,10, 0,08, 0,06, 0,04, 0,02, 0,01, 0,005, 0,001, 0,0005, 0,0001, 0,00005 или 0,00001% по отношению к детектируемому ответу для всех C5-углеводородов данной композиции. В некоторых вариантах изобретения, указанная композиция дает относительный детектируемый ответ для 1,3-циклопентадиена, цис-1,3-пентадиена, транс-1,3-пентадиена, 1-пентина, 2-пентина, 1-пентена, 2-метил-1-бутена, 3-метил-1-бутина, транс-пиперилена, цис-пиперилена, пент-4-ен-1-ина, транс-пент-3-ен-1-ина или цис-пент-3-ен-1-ина, который составляет по меньшей мере или примерно 0,12, 0,10, 0,08, 0,06, 0,04, 0,02, 0,01, 0,005, 0,001, 0,0005, 0,0001, 0,00005 или 0,00001% по отношению к детектируемому ответу для всех C5-углеводородов в данной композиции. В некоторых вариантах изобретения, композиция изопрена содержит примерно от 0,02 до 0,04%, примерно от 0,04 до 0,06%, примерно от 0,06 до 0,08%, примерно от 0,08 до 0,10%, или примерно от 0,10 до 0,12% C5-углеводородов, не являющихся изопреном (таких как 1,3-циклопентадиен, цис-1,3-пентадиен, транс-1,3-пентадиен, 1-пентин, 2-пентин, 1-пентен, 2-метил-1-бутен, 3-метил-1-бутин, транс-пиперилен, цис-пиперилен, пент-4-ен-1-ин, транс-пент-3-ен-1-ин или цис-пент-3-ен-1-ин), по общей массе всех C5-углеводородов в указанной композиции.
В некоторых вариантах изобретения, композиция изопрена содержит менее чем или примерно 50, 40, 30, 20, 10, 5, 1, 0,5, 0,1, 0,05, 0,01 или 0,005 мкг/л соединения, которое ингибирует полимеризацию изопрена для любого соединения в композиции, которое ингибирует полимеризацию изопрена. В некоторых вариантах изобретения, композиция изопрена содержит примерно от 0,005 до 50, например, примерно от 0,01 до 10, примерно от 0,01 до 5, примерно от 0,01 до 1, примерно от 0,01 до 0,5, или примерно от 0,01 до 0,005 мкг/л соединения, которое ингибирует полимеризацию изопрена для любого соединения в композиции, которое ингибирует полимеризацию изопрена. В некоторых вариантах изобретения, композиция изопрена содержит менее чем или примерно 50, 40, 30, 20, 10, 5, 1, 0,5, 0,1, 0,05, 0,01, или 0,005 мкг/л углеводородов, не являющихся изопреном (таких как 1,3-циклопентадиен, цис-1,3-пентадиен, транс-1,3-пентадиен, 1-пентин, 2-пентин, 1-пентен, 2-метил-1-бутен, 3-метил-1-бутин, транс-пиперилен, цис-пиперилен, пент-4-ен-1-ин, транс-пент-3-ен-1-ин или цис-пент-3-ен-1-ин). В некоторых вариантах изобретения, композиция изопрена содержит примерно от 0,005 до 50, например, примерно от 0,01 до 10, примерно от 0,01 до 5, примерно от 0,01 до 1, примерно от 0,01 до 0,5, или примерно от 0,01 до 0,005 мкг/л углеводорода, не являющегося изопреном. В некоторых вариантах изобретения, композиция изопрена включает менее чем или примерно 50, 40, 30, 20, 10, 5, 1, 0,5, 0,1, 0,05, 0,01 или 0,005 мкг/л белка или жирной кислоты (такой как белок или жирная кислота, которые обычно ассоциируются с натуральным каучуком).
В некоторых вариантах изобретения, композиция изопрена содержит менее чем или примерно 10, 5, 1, 0,8, 0,5, 0,1, 0,05, 0,01, или 0,005 м.д. альфа-ацетиленов, пипериленов, ацетонитрила или 1,3-циклопентадиена. В некоторых вариантах изобретения, композиция изопрена содержит менее чем или примерно 5, 1, 0,5, 0,1, 0,05, 0,01, или 0,005 м.д. серы или алленов. В некоторых вариантах изобретения, композиция изопрена содержит менее чем или примерно 30, 20, 15, 10, 5, 1, 0,5, 0,1, 0,05, 0,01 или 0,005 м.д. всех ацетиленов (таких как пентин-1, бутин-2, 2MB1-3-ин и 1-пентин-4-ин). В некоторых вариантах изобретения, композиция изопрена содержит менее чем или примерно 2000, 1000, 500, 200, 100, 50, 40, 30, 20, 10, 5, 1, 0,5, 0,1, 0,05, 0,01, или 0,005 м.д. димеров изопрена, таких как циклические димеры изопрена (например, циклические C10-соединения, образующиеся в результате димеризации двух изопреновых звеньев).
В некоторых вариантах изобретения, композиция изопрена включает этанол, ацетон, прениловый C5-спирт (такой как 3-метил-3-бутен-1-ол или 3-метил-2-бутен-1-ол), или любые два или более вышеупомянутых соединений. В конкретных вариантах изобретения, композиция изопрена содержит более чем или примерно 0,005, 0,01, 0,05, 0,1, 0,5, 1, 5, 10, 20, 30, 40, 60, 80, 100 или 120 мкг/л этанола, ацетона, пренилового C5-спирта (такого как 3-метил-3-бутен-1-ол или 3-метил-2-бутен-1-ол), или любых двух или более вышеупомянутых соединений. В некоторых вариантах изобретения, композиция изопрена содержит примерно от 0,005 до 120, например, примерно от 0,01 до 80, примерно от 0,01 до 60, примерно от 0,01 до 40, примерно от 0,01 до 30, примерно от 0,01 до 20, примерно от 0,01 до 10, примерно от 0,1 до 80, примерно от 0,1 до 60, примерно от 0,1 до 40, примерно от 5 до 80, примерно от 5 до 60, или примерно от 5 до 40 мкг/л этанола, ацетона, пренилового C5-спирта, или любых двух или более вышеупомянутых соединений.
В некоторых вариантах изобретения, композиция изопрена содержит одно или более из следующих компонентов, таких как 2-гептанон, 6-метил-5-гептен-2-он, 2,4,5-триметилпиридин, 2,3,5-триметилпиразин, цитронеллаль, ацетальдегид, метантиол, метилацетат, 1-пропанол, диацетил, 2-бутанон, 2-метил-3-бутен-2-ол, этилацетат, 2-метил-1-пропанол, 3-метил-1-бутаналя, 3-метил-2-бутанон, 1-бутанол, 2-пентанон, 3-метил-1-бутанол, этилизобутират, 3-метил-2-бутенaл, бутилацетат, 3-метилбутилацетат, 3-метил-3-бут-1-енилацетат, 3-метил-2-бут-1-енилацетат, (E)-3,7-диметил-1,3,6-октатриен, (Z)-3,7-диметил-1,3,6-октатриен и 2,3-циклогептенолпиридин, или линейный изопреновый полимер (такой как линейный изопреновый димер или линейный изопреновый тример, образующийся в результате полимеризации множества изопреновых звеньев). В различных вариантах изобретения, количество одного из этих компонентов по отношению к количеству изопрена в процентах по массе (то есть масса компонента, деленная на массу изопрена × 100) превышает или составляет примерно 0,01, 0,02, 0,05, 0,1, 0,5, 1, 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100 или 110% (масс./масс.). В некоторых вариантах изобретения, относительный детектируемый ответ для второго соединения по отношению к детектируемому ответу для изопрена составляет более чем или примерно 0,01, 0,02, 0,05, 0,1, 0,5, 1, 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100 или 110%. В различных вариантах изобретения, количество одного из этих компонентов по отношению к количеству изопрена в процентах по массе (то есть масса компонента, деленная на массу изопрена × 100) составляет примерно от 0,01 до 105% (масс./масс.), например, примерно от 0,01 до 90, примерно от 0,01 до 80, примерно от 0,01 до 50, примерно от 0,01 до 20, примерно от 0,01 до 10, примерно от 0,01 до 50, примерно от 0,01 до 50, примерно от 0,01 до 50, или от 0,1 до примерно 20% (масс./масс.).
В некоторых вариантах изобретения, композиция изопрена включает одно или более из следующих компонентов, таких как спирт, альдегид или кетон (например, любой из описанных здесь спиртов, альдегидов или кетонов). В некоторых вариантах изобретения, композиция изопрена включает (i) спирт и альдегид, (ii) спирт и кетон, (iii) альдегид и кетон или (iv) спирт, альдегид и кетон.
В некоторых вариантах изобретения, композиция изопрена включает один или более из следующих компонентов, таких как метанол, ацетальдегид, этанол, метантиол, 1-бутанол, 3-метил-1-пропанол, ацетон, уксусная кислота, 2-бутанон, 2-метил-1-бутанол или индол. В некоторых вариантах изобретения, композиция изопрена включает 1 м.д. одного или более из следующих компонентов: метанола, ацетальдегида, этанола, метантиола, 1-бутанола, 3-метил-1-пропанола, ацетона, уксусной кислоты, 2-бутанона, 2-метил-1-бутанола или индола. В некоторых вариантах изобретения, концентрация более чем одного или более из следующих компонентов, а именно, метанола, ацетальдегида, этанола, метантиола, 1-бутанола, 3-метил-1-пропанола, ацетона, уксусной кислоты, 2-бутанона, 2-метил-1-бутанола или индола составляет примерно 1 до 10000 м.д. в композиции изопрена (такой как отходящий газ перед очисткой). В некоторых вариантах изобретения, композиция изопрена (такая как отходящий газ после одной или более стадий очистки) включает один или более из следующих компонентов, таких как метанол, ацетальдегид, этанол, метантиол, 1-бутанол, 3-метил-1-пропанол, ацетон, уксусная кислота, 2-бутанон, 2-метил-1-бутанол или индол, в концентрации примерно от 1 до 100 м.д., например, примерно от 1 до 10 м.д., примерно от 10 до 20 м.д., примерно от 20 до 30 м.д., примерно от 30 до 40 м.д., примерно от 40 до 50 м.д., примерно от 50 до 60 м.д., примерно от 60 до 70 м.д., примерно от 70 до 80 м.д., примерно от 80 до 90 м.д., или примерно от 90 до 100 м.д. Летучие органические соединения клеточных культур (такие как летучие органические соединения в зоне «хэдспейс» клеточных культур) могут быть проанализированы стандартными методами, такими как методы, описанные в настоящей заявке, или другими стандартными методами, такими как масс-спектрометия с переносом протонов (см., например, публикацию Bunge et al., Applied and Environmental Microbiology, 74(7):2179-2186, 2008, которая во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание анализа летучих органических соединений).
В некоторых вариантах изобретения, указанная композиция содержит более чем примерно 2 мг изопрена, например, более чем или примерно 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 мг изопрена. В некоторых вариантах изобретения, указанная композиция содержит более чем примерно 2, 5, 10, 20, 30, 40, 50, 60, 70, 80, 90, 100 г изопрена. В некоторых вариантах изобретения, количество изопрена в данной композиции составляет примерно от 2 до 5000 мг, например, примерно от 2 до 100 мг, примерно от 100 до 500 мг, примерно от 100 до 1000 мг, примерно от 1000 до 2000 мг, или примерно от 2000 до 5000 мг. В некоторых вариантах изобретения, количество изопрена в данной композиции составляет примерно от 20 до 5,000 мг, примерно от 100 до 5000 мг, примерно от 200 до 2000 мг, примерно от 200 до 1000 мг, примерно от 300 до 1000 мг, или примерно от 400 до 1000 мг. В некоторых вариантах изобретения, более чем или примерно 20, 25, 30, 40, 50, 60, 70, 80, 90 или 95% по массе летучей органической фракции данной композиции составляет изопрен.
В некоторых вариантах изобретения, указанная композиция включает этанол. В некоторых вариантах изобретения, указанная композиция включает примерно от 75 до 90 масс.% этанола, например, примерно от 75 до 80%, примерно от 80 до 85%, или примерно от 85 до 90% масс.% этанола. В некоторых вариантах изобретения, указанная композиция включает примерно от 4 до 15 масс.% изопрена, например, примерно от 4 до 8%, примерно от 8 до 12%, или примерно от 12 до 15% масс.% изопрена.
В некоторых вариантах изобретения, клетка, содержащая одну или более гетерологичных нуклеиновых кислот, кодирующих полипептид изопренсинтазы, полипептид DXS, полипептид IDI и/или полипептид пути MVA, продуцирует изопреноидное соединение (такое как соединение с 10 или более атомами углерода, образующееся в результате реакции одной или более молекул IPP с одной или более молекулами DMAPP) в количестве, которое более чем или примерно в 2, 3, 5, 10, 25, 50, 100, 150, 200, 400 раз или более превышает количество изопреноидного соединения, продуцируемого из соответствующих клеток, культивируемых, в основном, в тех же условиях, но в отсутствии одной или более гетерологичных нуклеиновых кислот. В некоторых вариантах изобретения, клетка, содержащая одну или более гетерологичных нуклеиновых кислот, кодирующих полипептид изопренсинтазы, полипептид DXS, полипептид IDI и/или полипептид пути MVA, продуцирует прениловый С5-спирт (такой как 3-метил-3-бутен-1-ол или 3-метил-2-бутен-1-ол) в количестве, которое более чем или примерно в 2, 3, 5, 10, 25, 50, 100, 150, 200, 400 раз или более превышает количество пренилового С5-спирта, продуцируемого из соответствующих клеток, культивируемых, в основном, в тех же условиях, но в отсутствии одной или более гетерологичных нуклеиновых кислот.
Репрезентативные методы очистки изопрена
В некоторых вариантах изобретения, любой из описанных здесь способов также включает регенерацию изопрена. Так, например, изопрен, продуцированный с применением композиций и способов согласно изобретению, может быть регенерирован стандартными методами, такими как десорбционная обработка газов, разделение на мембране, фракционирование, адсорбция/десорбция, диффузионное испарение, тепловая и вакуумная десорбция изопрена из твердой фазы, или экстракция изопрена, иммобилизованного или адсорбированного на твердой фазе, растворителем (см., например, патенты США №№ 4703007 и 4570029, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание методов регенерации и очистки). В конкретных вариантах изобретения, для регенерации изопрена используется экстракционная перегонка со спиртом (таким как этанол, метанол, пропанол или их комбинации). В некоторых вариантах изобретения, регенерация изопрена включает выделение изопрена в жидкой форме (такой как чистый раствор изопрена или раствор изопрена в растворителе). Десорбционная обработка газов включает удаление паров изопрена из потока отходящего газа в процессе ферментации в непрерывной культуре. Такое удаление может быть достигнуто несколькими различными способами, включая, но не ограничиваясь ими, адсорбция на твердой фазе, распределение по всей жидкой фазе, или прямая конденсация (такая как конденсация, достигаемая с помощью охлаждающего змеевика или путем увеличения давления). В некоторых вариантах изобретения, обогащение мембраны потоком паров разбавленного изопрена при температуре выше точки росы данного пара, приводит к конденсации жидкого изопрена. В некоторых вариантах изобретения, изопрен подвергают сжатию и конденсации.
Регенерация изопрена может включать одну или более стадий. В некоторых вариантах изобретения, удаление паров изопрена из отходящих газов, образующихся при ферментации, и превращение изопрена в жидкую фазу осуществляют одновременно. Так, например, изопрен может быть непосредственно конденсирован из потока отходящего газа с образованием жидкости. В некоторых вариантах изобретения, удаление паров изопрена из отходящих газов, образующихся при ферментации, и превращение изопрена в жидкую фазу осуществляют последовательно. Так, например, изопрен может быть адсорбирован на твердой фазе, а затем экстрагирован из твердой фазы растворителем.
В некоторых вариантах изобретения, любые из описанных здесь способов также включают очистку изопрена. Так, например, изопрен, продуцируемый с применением композиций и способов согласно изобретению, может быть очищен стандартными методами. Термин «очистка» означает процесс отделения изопрена от одного или более компонентов, присутствующих при продуцировании изопрена. В некоторых вариантах изобретения, изопрен получают, в основном, в виде чистой жидкости. Примерами способов очистки являются (i) перегонка из раствора в жидком экстрагирующем растворителе и (ii) хроматография. Используемый здесь термин «очищенный изопрен» означает изопрен, который был отделен от одного или более компонентов, присутствующих при продуцировании изопрена. В некоторых вариантах изобретения, по меньшей мере примерно 20 масс.% изопрена не содержит других компонентов, присутствующих при продуцировании изопрена. В различных вариантах изобретения, изопрен имеет чистоту, составляющую по меньшей мере или примерно 25%, 30%, 40%, 50%, 60%, 70%, 75%, 80%, 90%, 95% или 99% по массе. Чистота может быть оценена любым подходящим методом, например, с помощью колоночной хроматографии, ВЭЖХ-анализа или ГХ-МС-анализа.
В некоторых вариантах изобретения, по меньшей мере часть газовой фазы, оставшуюся после проведения одной или более стадий регенерации для удаления изопрена, подвергают рециклингу путем введения газовой фазы в культуральную клеточную систему (такую как ферментер) для продуцирования изопрена.
В некоторых вариантах изобретения, любой из описанных здесь способов также включает полимеризацию изопрена. Так, например, для полимеризации очищенного изопрена с образованием цис-полиизопрена или других последующих продуктов могут быть применены стандартные методы. В соответствии с этим, настоящее изобретение также относится к шинам, содержащим полиизопрен, таким как цис-1,4-полиизопрен и/или транс-1,4-полиизопрен, полученный из любых описанных здесь композиций изопрена.
ПРИМЕРЫ
В примерах, которые приводятся лишь в целях иллюстрации, а поэтому не должны рассматриваться как ограничение настоящего изобретения, также подробно описаны и представлены обсуждаемые выше аспекты и варианты настоящего изобретения. Если это не оговорено особо, то температуры даны в градусах Цельсия, а давление является атмосферным или близким к атмосферному. Ранее описанные примеры и подробное описание приводится лишь в целях иллюстрации и не должно рассматриваться как ограничение изобретения. Все публикации, патентные заявки и патенты, цитируемые в настоящей заявке, вводятся в настоящее описание посредством ссылки так, как если бы каждая отдельная публикация, патентная заявка или патент были конкретно и отдельно введены посредством ссылки. В частности, все цитируемые здесь публикации точно вводятся в настоящее изобретение посредством ссылки в целях описания и обсуждения композиций и методов, которые могут быть применены в соответствии с настоящим изобретением. Хотя изложенное выше изобретение проиллюстрировано на отдельных деталях, которые, для лучшего понимания изобретения, приведены в графическом материале и в примерах, однако, совершенно очевидно, что, исходя из описания настоящего изобретения, средний специалист в данной области может внести некоторые изменения и модификации, не выходящие за рамки существа и объема прилагаемой формулы изобретения.
ПРИМЕР 1: Продуцирование изопрена в E.coli, экспрессирующей рекомбинантную изопренсинтазу пуэрарии
I. Конструирование векторов для экспрессии изопренсинтазы пуэрарии в E.coli
Последовательность белка пуэрарии (Pueraria montana), кодируемую геном изопренсинтазы (IspS), получали из банка генов GenBank (AAQ84170). Ген изопренсинтазы пуэрарии, оптимизированный по встречаемости кодонов E.coli, закупали у DNA2.0 (SEQ ID NO: 1). Ген изопренсинтазы выделяли из закупленной плазмиды путем гидролиза рестриктирующими эндонуклеазами BspLU11I/PstI, а затем подвергали гель-очистке и лигировали в плазмиду pTrcHis2B (Invitrogen), которую гидролизовали ферментами NcoI/PstI. Эту конструкцию создавали так, чтобы стоп-кодон в гене изопренсинтазы находился со стороны 5'-конца по отношению к PstI-сайту. В результате, при экспрессии данной конструкции, His-метка не была присоединена к белку изопренсинтазы. Полученную плазмиду pTrcKudzu подтверждали путем секвенирования (фигуры 2 и 3).
Ген изопренсинтазы также клонировали в pET16b (Novagen). В этом случае, ген изопренсинтазы встраивали в pET16b так, чтобы рекомбинантный белок изопренсинтазы содержал N-концевую His-метку. Ген изопренсинтазы амплифицировали из pTrcKudzu с помощью ПЦР с использованием набора праймеров pET-His-Kudzu-2F: 5'-CGTGAGATCATATGTGTGCGACCTCTTCTCAATTTAC (SEQ ID NO:3) и pET-His-Kudzu-R: 5'-CGGTCGACGGATCCCTGCAGTTAGACATACATCAGCTG (SEQ ID NO:4). Эти праймеры присоединяли NdeI-сайт у 5'-конца и BamH1-сайт у 3'-конца гена, соответственно. Плазмиду pTrcKudzu, описанную выше, использовали в качестве ДНК-матрицы, ДНК-полимеразу HERCULASE (Stratagene) использовали в соответствии с инструкциями производителей, а праймеры добавляли в концентрации 10 пM. ПЦР осуществляли в общем объеме 25 мкл. ПЦР-продукт гидролизовали ферментами NdeI/BamH1 и клонировали в вектор pET16b, гидролизованный теми же самыми ферментами. Смесь для лигирования переносили в клетки E.coli Top10 (Invitrogen), и «правильный» клон отбирали путем секвенирования. Затем полученную плазмиду, в которой ген изопренсинтазы пуэрарии экспрессировался с промотора T7, обозначали pETNHisKudzu (фигуры 4 и 5).
Ген изопренсинтазы пуэрарии также клонировали в низкокопийную плазмиду pCL1920. Для амплификации гена изопренсинтазы пуэрарии из pTrcKudzu, описанной выше, использовали праймеры. Прямой праймер добавлял HindIII-сайт и консенсусную последовательность RBS E.coli к 5'-концу. Клонирующий PstI-сайт уже присутствовал в pTrcKudzu непосредственно у 3'-конца стоп-кодона, а поэтому обратный праймер был сконструирован так, чтобы конечный ПЦР-продукт включал PstI-сайт. Указанные праймеры имели следующие последовательности: ΗindIII-rbs-Kudzu F (прямой): 5'-CATATGAAAGCTTGTATCGATTAAATAAGGAGGAATAAACC (SEQ ID NO:5) и BamH1-Kudzu R (обратный): 5'-CGGTCGACGGATCCCTGCAGTTAGACATACATCAGCTG (SEQ ID NO:4). ПЦР-продукт амплифицировали с использованием полимеразы ΗERCULASE (Stratagene), а также праймеров в концентрации 10 пM и 1 нг матричной ДНК (pTrcKudzu). Протокол амплификации включает 30 циклов (95°C, 1 минута, 60°C, 1 минута, 72°C, 2 минуты). Продукт гидролизовали ферментами HindIII и PstI и лигировали в плазмиду pCL1920, которую также гидролизовали ферментами HindIII и PstI. Смесь для лигирования переносили в клетки E. coli Top10. Несколько трансформантов подтверждали путем анализа последовательности. Полученную плазмиду обозначали pCL-lac-Kudzu (фигуры 6 и 7).
II. Определение уровня продуцирования изопрена
Для культур в шейкерных колбах, 1 мл культуры переносили из шейкерных колб в 20-миллилитровые сосуды CTC с зоной «хэдспейс» (сосуд Agilent, номер по каталогу 5188 2753, и крышка, номер по каталогу 5188 2759). Крышку плотно завинчивали, и сосуды инкубировали при эквивалентной температуре со встряхиванием при 250 об/мин. Через 30 минут, сосуды вынимали из инкубатора и анализировали, как описано ниже (см. таблицу 1, где указаны некоторые экспериментальные величины данного анализа).
В случаях, когда продуцирование изопрена в ферментерах подтверждалось, из ферментера брали образцы отходящего газа и сразу анализировали, как описано ниже (см. таблицу 2, где указаны некоторые экспериментальные величины данного анализа).
Этот анализ осуществляли с использованием ГХ/МС-системы Agilent 6890, подсоединенной к автоматическому устройству для взятия образцов CTC Analytics CombiPAL (Switzerland), работающему в режиме «хэдспейс». Для разделения аналитов использовали ГХ/МС-колонку Agilent HP-5MS (30 м × 0,25 мм; пленка толщиной 0,25 мкм). Устройство для взятия образцов устанавливали на «впрыск» 500 мкл газа в зону «хэдспейс». В ГХ/МС-методе, в качестве газа-носителя использовали гелий при скорости потока 1 мл/мин. Входное отверстие для впрыска поддерживали при температуре 250°C с отношением деления потока 50:1. Во время анализа, продолжающегося 2 минуты, температуру печи поддерживали при 37°С. Селективный масс-детектор Agilent 5793N работает в режиме мониторинга одного иона (SIM) при отношении масса/заряд (m/z) 67. Для элюирования постоянных газов, детектор отключали на 1,4-1,7 минут. В этих условиях было установлено, что изопрен (2-метил-1,3-бутадиен) элюировался через 1,78 минут. Для количественной оценки абсолютного количества изопрена использовали калибровочную таблицу, которая показывала линейную зависимость на участке от 1 мкг/л до 2000 мкг/л. По оценкам, проводимым этим методом, предел детектирования составлял от 50 до 100 нг/л.
III. Продуцирование изопрена в шейкерных колбах, содержащих клетки E.coli, экспрессирующие рекомбинантную изопренсинтазу
Векторы, описанные выше, вводили в штамм E. coli BL21 (Novagen) для продуцирования штаммов BL21/ptrcKudzu, BL21/pCL-lac-Kudzu и BL21/pETHisKudzu. Штаммы для выделения распределяли на агаре LA (агаре Луриа), содержащем карбенициллин (50 мкг/мл) и инкубировали в течение ночи при 37°C. Моноколонии инокулировали в 250-миллилитровые шейкерные колбы с перегородками, содержащие 20 мл бульона Луриа-Бертани (LB) и карбенициллин (100 мкг/мл). Культуры выращивали в течение ночи при 20°C со встряхиванием при 200 об/мин. OD600 ночных культур измеряли, а затем культуры разводили в 250-миллилитровой шейкерной колбе с перегородкой, содержащей 30 мл среды MagicMedia (Invitrogen) + карбенициллин (100 мкг/мл) до OD600 ~0,05. Эту культуру инкубировали при 30°C со встряхиванием при 200 об/мин. Когда OD600 составляла ~0,5-0,8 добавляли 400 мкМ IPTG, и клетки инкубировали еще 6 часов при 30°С со встряхиванием при 200 об/мин. Через 0, 2, 4 и 6 часов после IPTG-индуцирования, 1 мл аликвот культур собирали, определяли OD600, и количество продуцированного изопрена измеряли, как описано выше. Результаты приводятся на фигуре 8.
IV. Продуцирование изопрена из BL21/ptrcKudzu в 14-литровом ферментере
Крупномасштабное продуцирование изопрена клетками E.coli, содержащими рекомбинантный ген изопренсинтазы пуэрарии, осуществляли путем их культивирования в периодической культуре с подпиткой. Среда для ферментации (TM2) содержала нижеследующие компоненты на литр: 13,6 г K2HPO4, 13,6 г KН2PO4, 2 г МgSO4 · 7 Н2О, 2 г моногидрата лимонной кислоты, 0,3 г цитрата железа(3)-аммония, 3,2 г (NН4)2SO4, 5 г дрожжевого экстракта, 1 мл 1000 × модифицированного раствора следовых количеств металла. Все компоненты добавляли одновременно и растворяли в diH2O. pH доводили до 6,8 добавлением гидроксида калия (KOH) и объем доводили до нужной величины. Конечный продукт стерилизовали только на 0,22 мкМ-фильтре (не обрабатывали в автоклаве). 1000 × модифицированный раствор следовых количеств металла имел следующий состав: 40 г лимонной кислоты · Н2О; 30 г МnSO4 · Н2О; 10 г NaCl, 1 г FeSO4 · 7 Н2О; 1 г CoCl2 · 6 Н2О, 1 г ZnSO · 7 Н2О, 100 мг CuSO4 · 5 Н2О, 100 мг H3ВО3, 100 мг NaMoO4 · 2 Н2О. Каждый компонент сразу растворяли в diН2О, рН доводили до 3,0 добавлением HCl/NaOH, а затем объем доводили до нужной величины и стерилизовали на фильтре 0,22 микрон.
Этот эксперимент осуществляли в 14-литровом биореакторе для мониторинга образования изопрена из глюкозы в нужном режиме ферментации, при pH 6,7 и при температуре 34°C. Инокулят штамма BL21/ptrcKudzu E.coli, взятый из замороженного сосуда, получали в среде, содержащей соевый пептон-дрожжевой экстракт-глюкозу. После достижения инокулятом OD550=0,6, две 600-миллилитровые колбы центрифугировали, и содержимое ресуспендировали в 70 мл супернатанта для переноса клеточного осадка (70 мл материала с OD 3,1) в биореактор. Через различные периоды времени после инокуляции брали образцы и определяли количество продуцированного изопрена, как описано выше. Результаты приводятся на фигуре 9.
ПРИМЕР 2: Продуцирование изопрена в E.coli, экспрессирующей рекомбинантную изопренсинтазу тополя
Последовательность белка изопренсинтазы тополя (Populus alba x Populus tremula) (Schnitzler, J-P, et al. (2005) Planta 222:777-786) получали из банка генов GenBank (CAC35696). Ген, оптимизированный по кодонам для E.coli, закупали у DNA2.0 (p9796-POP1ar, фигуры 30 и 31). Ген изопренсинтазы выделяли из закупленной плазмиды путем гидролиза рестриктирующими эндонуклеазами BspLU11I/PstI, а затем подвергали гель-очистке и лигировали в плазмиду pTrcHis2B, которую гидролизовали ферментами NcoI/PstI. Эту конструкцию клонировали так, чтобы стоп-кодон во вставке находился перед PstI-сайтом, в результате чего получали конструкцию, в которой His-метка не была присоединена к белку изопренсинтазы. Полученную плазмиду pTrcPOP1ar (фигуры 32 и 33) подтверждали путем секвенирования.
ПРИМЕР 3: Продуцирование изопрена в Panteoa citrea, экспрессирующей рекомбинантную изопренсинтазу пуэрарии
Плазмиды pTrcKudzu и pCL-lac-Kudzu, описанные в примере 1, подвергали электропорации в P. citrea (патент США No. 7241587). Трансформанты отбирали на среде LA, содержащей карбенициллин (200 мкг/мл) или спектиномицин (50 мкг/мл), соответственно. Продуцирование изопрена в шейкерных колбах и определение количества продуцируемого изопрена осуществляли, как описано в примере 1 для штаммов E.coli, экспрессирующих рекомбинантную изопренсинтазу пуэрарии. Результаты приводятся на фигуре 10.
ПРИМЕР 4: Продуцирование изопрена в Bacillus subtilis, экспрессирующей рекомбинантную изопренсинтазу пуэрарии
I. Конструирование плазмиды, реплицирующейся в B. subtilis, для экспрессии изопренсинтазы пуэрарии
Экспрессию гена изопренсинтазы пуэрарии осуществляли в штамме aprEnprE Pxyl-comK (BG3594comK) Bacillus subtilis с использованием реплицирующейся плазмиды (pBS19 с кластером резистентности к хлорамфениколу) под контролем промотора aprE. Ген изопренсинтазы, промотор aprE и терминатор транскрипции амплифицировали по отдельности и лигировали с помощью ПЦР. Затем эту конструкцию клонировали в pBS19 и переносили в B. subtilis.
a) Амплификация промотора aprE
Промотор aprE амплифицировали из хромосомной ДНК Bacillus subtilis с использованием нижеследующих праймеров:
CF 797 (+) старт-кодон - промотор aprE-MfeI
5'- GACATCAATTGCTCCATTTTCTTCTGCTATC (SEQ ID NO:58)
CF 07-43 (-) лигирование промотора aprE с ispS пуэрарии
5'- ATTGAGAAGAGGTCGCACACACTCTTTACCCTCTCCTTTTA (SEQ ID NO:59)
b) Амплификация гена изопренсинтазы
Ген изопренсинтазы пуэрарии амплифицировали из плазмиды pTrcKudzu (SEQ ID NO:2). Ген был оптимизирован по кодонам для E.coli и синтезирован в DNA 2.0. При этом, использовали нижеследующие праймеры:
CF 07-42 (+) лигирование промотора aprE с геном изопренсинтазы пуэрарии (старт-кодон GTG) 5'- TAAAAGGAGAGGGTAAAGAGTGTGTGCGACCTCTTCTCAAT (SEQ ID NO:60)
CF 07-45 (-) лигирование 3'-конца гена изопренсинтазы пуэрарии с терминатором
5'- CCAAGGCCGGTTTTTTTTAGACATACATCAGCTGGTTAATC (SEQ ID NO:61)
c) Амплификация терминатора транскрипции
Терминатор от гена щелочной сериновой протеазы Bacillus amyliquefaciens амплифицировали из предварительно секвенированной плазмиды pJHPms382 с использованием нижеследующих праймеров:
CF 07-44 (+) лигирование 3'-конца гена изопренсинтазы пуэрарии с терминатором
5'- GATTAACCAGCTGATGTATGTCTAAAAAAAACCGGCCTTGG (SEQ ID NO:62)
CF 07-46 (-) Конец терминатора B. amyliquefaciens (BamHI)
5'- GACATGACGGATCCGATTACGAATGCCGTCTC (SEQ ID NO:63)
Фрагмент пуэрарии лигировали с фрагментом терминатора с помощью ПЦР с использованием нижеследующих праймеров:
CF 07-42 (+)лигирование промотора aprE с геном изопренсинтазы пуэрарии (старт-кодон GTG) 5'-TAAAAGGAGAGGGTAAAGAGTGTGTGCGACCTCTTCTCAAT (SEQ ID NO:61)
CF 07-46 (-) Конец терминатора B. amyliquefaciens (BamHI)
5'- GACATGACGGATCCGATTACGAATGCCGTCTC (SEQ ID NO:63)
Фрагмент терминатора пуэрарии лигировали с фрагментом промотора с помощью ПЦР с использованием нижеследующих праймеров:
CF 797 старт-кодон - промотор aprE-MfeI
5'- GACATCAATTGCTCCATTTTCTTCTGCTATC (SEQ ID NO:64)
CF 07-46 (-) конец терминатора B. amyliquefaciens (BamHI)
5'- GACATGACGGATCCGATTACGAATGCCGTCTC (SEQ ID NO:63)
Гибридный ПЦР-фрагмент очищали с помощью набора Qiagen и гидролизовали рестриктирующими ферментами MfeI и BamHI. Этот гидролизованный ДНК-фрагмент подвергали гель-очистке с помощью набора Qiagen и лигировали с вектором, известным как pBS19, который был гидролизован ферментами EcoRI и BamHI и подвергнут гель-очистке.
Смесь для лигирования переносили в клетки Тор10 E.coli, и колонии отбирали на планшетах с LA+50 карбенициллина. Было отобрано всего шесть колоний, и эти колонии культивировали в течение ночи в LB+50 карбенициллина, а затем плазмиды выделяли с помощью набора Qiagen. Плазмиды гидролизовали ферментами EcoRI и BamHI для анализа на вставку и три «правильных» плазмиды отдавали на секвенирование, проводимое с использованием нижеследующих праймеров:
CF 149 (+) EcoRI старт-кодон промотора aprE
5'-GACATGAATTCCTCCATTTTCTTCTGC (SEQ ID NO:65)
CF 847 (+) последовательность в pXX 049 (конец промотора aprE) 5'-AGGAGAGGGTAAAGAGTGAG (SEQ ID NO:66)
CF 07-45 (-) Лигирование 3'-конца изопренсинтазы пуэрарии с терминатором
5'-CCAAGGCCGGTTTTTTTTAGACATACATCAGCTGGTTAATC (SEQ ID NO:61)
CF 07-48 (+) Секвенирующий праймер для изопренсинтазы пуэрарии 5'-CTTTTCCATCACCCACCTGAAG (SEQ ID NO:67)
CF 07-49 (+) Секвенирование изопренсинтазы пуэрарии
5'-GGCGAAATGGTCCAACAACAAAATTATC (SEQ ID NO:68)
Плазмиду, обозначенную pBS Kudzu #2 (фигуры 52 и 12), подтверждали путем секвенирования и переносили в штамм-хозяин BG 3594 comK, Bacillus subtilis. Отбор проводили на планшетах с LA+5 хлорамфеникола. Трансформанты отбирали и переносили в моноколонии на LA+5 хлорамфеникола, в затем культивировали в LB+5 хлорамфеникола до тех пор, пока OD600 не достигала 1,5. Трансформанты хранили в замороженном виде в сосудах при -80°C в присутствии глицерина. Полученный штамм обозначали CF443.
II. Продуцирование изопрена в шейкерных колбах, содержащих клетки B. subtilis, экспрессирующие рекомбинантную изопренсинтазу
Ночные культуры инокулировали моноколонией CF 443, взятой из среды LA + хлорамфеникол (Cm, 25 мкг/мл). Культуры выращивали в LB + Cm при 37°C со встряхиванием при 200 об/мин. Эти ночные культуры (1 мл) использовали для инокуляции 250-миллилитровых шейкерных колб с перегородкой, содержащих 25 мл среды Grants II и хлорамфеникол в конечной концентрации 25 мкг/мл. Среда Grants II имела следующий состав: 10 г соевого пептона (Soytone), 3 мл 1M K2HPO4, 75 г глюкозы, 3,6 г мочевины, 100 мл 10 × MOPS, дост. кол. до 1 л получали путем добавления H2O, pH 7,2; среда 10 × MOPS имела следующий состав: 83,72 г MOPS, 7,17 г трицина, 12 г таблеток KOH, 10 мл 0,276M раствора K2SO4, 10 мл 0,528M раствора MgCl2, 29,22 г NaCl, 100 мл 100 × микроэлементов; дост. кол. до 1 л получали путем добавления H2O; а среда 100 × микроэлементов имела состав: 1,47 г CaCl2 · 2H2O, 0,4 г FeSO4 · 7H2О, 0,1 г MnSO4 · H2О, 0,1 г ZnSO4 · H2O, 0,05 г CuCl2 · 2H2O, 0,1 г CoCl2 · 6H2O, 0,1 г Na2MoO4 · 2H2O, дост. кол. до 1 л получали путем добавления H2O. Шейкерные колбы инкубировали при 37°С, и образцы брали через 18, 24 и 44 часов. Через 18 часов брали образцы из зоны «хэдспейс» штамма CF443 и контрольного штамма. За эти 18 часов происходила аккумуляция изопрена. Количество изопрена определяли с помощью газовой хроматографии, как описано в примере 1. Продуцирование изопрена значительно усиливалось при экспрессии рекомбинантной изопренсинтазы (фигура 11).
III. Продуцирование изопрена штаммом CF443 в 14-литровом ферментере
Крупномасштабное продуцирование изопрена клетками B. subtilis, содержащими рекомбинантный ген изопренсинтазы пуэрарии на реплицирующейся плазмиде, определяли в периодической культуре с подпиткой. Штамм Bacillus CF 443, экспрессирующий ген изопренсинтазы пуэрарии, или контрольный штамм, который не экспрессировал гена изопренсинтазы пуэрарии, культивировали путем стандартной ферментации в периодической культуре с подпиткой в питательной среде, содержащей соевую муку (Cargill), фосфат натрия и калия, сульфат магния и раствор лимонной кислоты, хлорида железа(3) и хлорида марганца. Перед ферментацией, среду подвергали мацерации в течение 90 минут с использованием смеси ферментов, включающей целлюлазы, гемицеллюлазы и пектиназы (см., WO95/04134). В 14-литровый ферментер с периодической культурой подавали 60% масс. глюкозы (декстрозы Cargill DE99, зеленого красителя ADM Versadex или инвертированного сахара Danisco) и 99% масс. масла (соевого масла Western Family, концентрация которого перед добавлением в культуральную клеточную среду составляла 99% масс.). Подпитку начинали после того, как глюкоза в периодической культуре не детектировалась. Скорость подпитки быстро меняли в течение нескольких часов и корректировали добавлением масла с равным количеством углерода. pH поддерживали при 6,8-7,4 с использованием 28% масс./об. гидроксида аммония. В случае образования пены, в среду добавляли противовспенивающий агент. Температуру ферментации поддерживали при 37°С, и сбраживаемую культуру встряхивали при 750 об/мин. В течение всего процесса ферментации проводили мониторинг различных других параметров, таких как pH, DO%, поток воздуха и давление. DO% поддерживали выше 20. Образцы брали в течение 36 часов и анализировали на рост клеток (OD550) и продуцирование изопрена. Результаты этих экспериментов представлены на фигурах 53A и 53B.
IV. Интеграция гена изопренсинтазы пуэрарии (ispS) в B. subtilis.
Ген изопренсинтазы пуэрарии клонировали в интегрирующуюся плазмиду (pJH101-cmpR) под контролем промотора aprE. В условиях данного теста изопрен не детектировался.
ПРИМЕР 5: Продуцирование изопрена в Trichoderma
I. Конструирование векторов для экспрессии изопренсинтазы пуэрарии в Trichoderma reesei
Ген IS пуэрарии, оптимизированный по кодонам Yarrowia lipolytica, синтезировали с помощью DNA 2.0 (SEQ ID NO:8) (фигура 13). Эта плазмида служила в качестве матрицы для дальнейшей реакции ПЦР-амплификации, включающей: 1 мкл плазмидной матрицы (20 нг/мкл), 1 мкл праймера EL-945 (10 мкM) 5'-GCTTATGGATCCTCTAGACTATTACACGTACATCAATTGG (SEQ ID NO:9), 1 мкл праймера EL-965 (10 мкM) 5'-CACCATGTGTGCAACCTCCTCCCAGTTTAC (SEQ ID NO: 10), 1 мкл dNTP (10 мM), 5 мкл 10x буфера для ДНК-полимеразы PfuUltra II Fusion HS, 1 мкл ДНК-полимеразы PfuUltra II Fusion HS, 40 мкл воды в общем реакционном объеме 50 мкл. Прямой праймер содержал дополнительные 4 нуклеотида у 5'-конца, которые не соответствовали гену изопренсинтазы пуэрарии, оптимизированному по кодонам Y. lipolytica, но которые были встроены для клонирования в вектор pENTR/D-TOPO. Обратный праймер содержал дополнительный 21 нуклеотид у 5'-конца, которые не соответствовали гену изопренсинтазы пуэрарии, оптимизированному по кодонам Y. lipolytica, но которые были встроены для клонирования в остовы других векторов. ПЦР-реакцию проводили с использованием термоячейки MJ Research PTC-200 в следующих условиях: 95°C, 2 минуты (только первый цикл), 95°C, 30 секунд, 55°C, 30 секунд, 72°C, 30 секунд (повторяли в 27 циклах), 72°C, 1 минута после последнего цикла. ПЦР-продукт анализировали на 1,2% E-геле для подтверждения успешной амплификации гена изопренсинтазы пуэрарии, оптимизированной по кодонам Y. lipolytica.
Затем ПЦР-продукт клонировали с использованием набора для клонирования TOPO pENTR/D-TOPO в соответствии с протоколом, рекомендованным производителем: 1 мкл смеси для ПЦР-реакции, 1 мкл солевого раствора, 1 мкл вектора TOPO pENTR/D-TOPO и 3 мкл воды в общем реакционном объеме 6 мкл. Реакционную смесь инкубировали при комнатной температуре в течение 5 минут. Один микролитр реакционной смеси TOPO переносили в химически компетентные клетки E.coli TOP10. Трансформанты отбирали на планшетах с LA+50 мкг/мл канамицина. Несколько колоний собирали, и каждую из них инокулировали в 5 мл-пробирку, содержащую среду LB+50 мкг/мл канамицина, и культуры выращивали в течение ночи при 37°С со встряхиванием при 200 об/мин. Плазмиды выделяли из пробирок с ночной культурой с использованием набора QIAprep Spin Miniprep в соответствии с протоколом производителей. Несколько плазмид секвенировали для подтверждения «правильной» последовательности ДНК.
Одну плазмиду pENTR/D-TOPO, кодирующую ген изопренсинтазы пуэрарии, оптимизированный по кодонам Y. lipolytica, использовали для клонирования методом Gateway в специально изготовленном векторе pTrex3g. Конструкция pTrex3g описана в WO 2005/001036 A2. Реакцию осуществляли в соответствии с протоколом производителей, прилагаемым к набору Gateway LR Clonase II Enzyme Mix (Invitrogen), содержащему 1 мкл донорного вектора pENTR/D-TOPO, включающего ген изопренсинтазы пуэрарии, оптимизированный по кодонам Y. lipolytica, 1 мкл целевого вектора pTrex3g, 6 мкл буфера TE, pH 8,0, в общем реакционном объеме 8 мкл. Реакционную смесь инкубировали при комнатной температуре в течение 1 часа, а затем добавляли 1 мкл раствора протеиназы К и инкубирование продолжали при 37°С в течение 10 минут. Затем 1 мкл реакционной смеси переносили в химически компетентные клетки E.coli TOP10. Трансформанты отбирали на планшетах с LA+50 мкг/мл карбенициллина. Несколько колоний собирали, и каждую из них инокулировали в 5 мл-пробирку, содержащую среду LB+50 мкг/мл карбенициллина, и культуры выращивали в течение ночи при 37°С со встряхиванием при 200 об/мин. Плазмиды выделяли из пробирок с ночной культурой с использованием набора QIAprep Spin Miniprep (Qiagen, Inc.) в соответствии с протоколом производителей. Несколько плазмид секвенировали для подтверждения «правильной» последовательности ДНК.
Биолистический перенос плазмиды pTrex3g, содержащей ген изопренсинтазы пуэрарии, оптимизированный по кодонам Y. lipolytica (фигура 14), в штамм Trichoderma reesei с четырьмя делециями осуществляли с использованием биолистической системы доставки частиц PDS-1000/HE (см. WO 2005/001036 A2). Выделение стабильных трансформантов и их оценку в шейкерных колбах осуществляли в соответствии с протоколом, описанным в примере 11 патентной публикации WO 2005/001036 A2.
II. Продуцирование изопрена в рекомбинантных штаммах T. reesei
1 миллилитр 15- и 36-часовых культур трансформантов изопренсинтазы, описанных выше, переносили в сосуды с зоной «хэдспейс». Затем сосуды герметично закрывали и инкубировали в течение 5 часов при 30°С. Затем измеряли уровень газа в зоне «хэдспейс» и изопрен идентифицировали методом, описанным в примере 1. Два трансформанта обнаруживали следовые количества изопрена. Количество изопрена может быть увеличено при инкубировании в течение 14 часов. В течение 14-часового инкубирования, два позитивных образца обнаруживали изопрен на уровне примерно 0,5 мкг/л. Нетрансформированный контроль не обнаруживал детектируемых уровней изопрена. Этот эксперимент показал, что T. reesei обладает способностью продуцировать изопрен из эндогенного предшественника при подаче экзогенной изопренсинтазы.
ПРИМЕР 6: Продуцирование изопрена в Yarrowia
I. Конструирование векторов для экспрессии изопренсинтазы пуэрарии в Yarrowia lipolytica
В качестве исходного материала для конструирования векторов, экспрессирующих ген изопренсинтазы пуэрарии в Yarrowia lipolytica, служил вектор pSPZ1(MAP29Spb). Полноразмерная последовательность этого вектора (SEQ ID No:11) представлена на фигуре 15.
С помощью ПЦР и с использованием хромосомной ДНК штамма GICC 120285 Y. lipolytica в качестве матрицы были амплифицированы нижеследующие фрагменты: ген URA3, не содержащий промотор, фрагмент гена рибосомной РНК 18S, терминатор транскрипции гена XPR2 Y. lipolytica и два фрагмента ДНК, содержащих промоторы генов XPR2 и ICL1. Были использованы нижеследующие ПЦР-праймеры:
ICL1 3
5'-GGTGAATTCAGTCTACTGGGGATTCCCAAATCTATATATACTGCAGGTGAC
(SEQ ID NO:69)
ICL1 5
5'-GCAGGTGGGAAACTATGCACTCC (SEQ ID NO:70)
XPR 3
5'-CCTGAATTCTGTTGGATTGGAGGATTGGATAGTGGG (SEQ ID NO:71)
XPR 5
5'-GGTGTCGACGTACGGTCGAGCTTATTGACC (SEQ ID NO:72)
XPRT3
5'- GGTGGGCCCGCATTTTGCCACCTACAAGCCAG (SEQ ID NO:73)
XPRT 5
5'-GGTGAATTCTAGAGGATCCCAACGCTGTTGCCTACAACGG (SEQ ID NO:74)
Y18S3
5'-GGTGCGGCCGCTGTCTGGACCTGGTGAGTTTCCCCG (SEQ ID NO:75)
Y18S 5
5'-GGTGGGCCCATTAAATCAGTTATCGTTTATTTGATAG (SEQ ID NO:76)
YURA3
5'-GGTGACCAGCAAGTCCATGGGTGGTTTGATCATGG (SEQ ID NO:77)
YURA 50
5'-GGTGCGGCCGCCTTTGGAGTACGACTCCAACTATG (SEQ ID NO:78)
YURA 51
5'-GCGGCCGCAGACTAAATTTATTTCAGTCTCC (SEQ ID NO:79)
Для ПЦР-амплификации использовали полимеразу PfuUltraII (Stratagene), закупаемые у поставщиков буфер и dNTP, 2,5 мкM праймеры и указанную матричную ДНК в соответствии с инструкциями производителей. Для амплификации проводили следующий цикл: 95°C, 1 минута; 34x (95°С, 30 секунд; 55°С, 30 секунд; 72°С, 3 минуты) и 10 минут при 72°С, а затем инкубирование при 4°С.
Молекулы синтетических ДНК, кодирующие ген изопренсинтазы пуэрарии, оптимизированный по кодонам для экспрессии в Yarrowia, получали от DNA 2.0 (фигура 16; SEQ ID NO: 12). Подробная схема конструирования плазмид pYLA(KZ1) и pYLI(KZ1), несущих синтетический ген изопренсинтазы пуэрарии, находящийся под контролем промоторов XPR2 и ICL1, соответственно, представлена на фигуре 18. Также были сконструированы контрольные плазмиды, в которых вместо гена изопренсинтазы был встроен ген фактора скрещивания (MAP29) (фигуры 18E и l8F).
Аналогичная процедура клонирования может быть проведена для экспрессии гена изопренсинтазы тополя (Populus alba x Populus tremula). Последовательность изопренсинтазы тополя описана в публикации Miller B. et al. (2001) Planta 213, 483-487 и представлена на фигуре 17 (SEQ ID NO: 13). Схема конструирования плазмид pYLA(POP1) и pYLI(POP1), несущих синтетический ген изопренсинтазы тополя, находящийся под контролем промоторов XPR2 и ICL1, соответственно, представлена на фигурах 18A и B.
II. Продуцирование изопрена рекомбинантными штаммами Y. lipolytica
Векторы pYLA(KZ1), pYLI(KZ1), pYLA(MAP29) и pYLI(MAP29) гидролизовали ферментом SacII и использовали для трансформации штамма CLIB 122 Y. lipolytica с применением стандартной процедуры сообщения уридиновой прототрофии, проводимой с использованием ацетата лития/полиэтиленгликоля. Вкратце, дрожжевые клетки, культивируемые в YEPD (1% дрожжевой экстракт, 2% пептон, 2% глюкоза) в течение ночи, собирали путем центрифугирования (4000 об/мин, 10 минут), один раз промывали стерильной водой и суспендировали в 0,1 M ацетата лития, pH 6,0. 200 мкл аликвот клеточной суспензии смешивали с раствором линеаризованной плазмидной ДНК (10-20 мкг), инкубировали в течение 10 минут при комнатной температуре и смешивали с 1 мл 50% ПЭГ 4000 в том же самом буфере. Затем суспензии инкубировали в течение 1 часа при комнатной температуре, после чего подвергали тепловому шоку в течение 2 минут при 42°C. Затем клетки высевали на SC-планшеты с his и leu (0,67% азотистого основания дрожжей, 2% глюкоза, 100 мг/л лейцина и 100 мг/л гистидина). Трансформанты обнаруживались после 3-4-дневного инкубирования при 30°С.
Три изолята, полученных после трансформации pYLA(KZ1), три изолята, полученных после трансформации pYLI(KZ1), два изолята, полученных после трансформации pYLA(MAP29), и два изолята, полученных после трансформации pYLI(MAP29), культивировали в течение 24 часов в среде YEP7 (1% дрожжевой экстракт, 2% пептон, pH 7,0) при 30°С со встряхиванием. Клетки от 10 мл культуры собирали центрифугированием, ресуспендировали в 3 мл свежей среды YEP7 и помещали в 15-миллилитровые сосуды с завинчивающейся крышкой. Сосуды инкубировали в течение ночи при комнатной температуре с легким встряхиванием (60 об/мин). Содержание изопрена в зоне «хэдспейс» этих сосудов анализировали с помощью газовой хроматографии с использованием масс-спектрометрического детектора, описанного в примере 1. Все трансформанты, полученные с использованием pYLA(KZ1) и pYLI(KZ1), продуцировали легко детектируемые количества изопрена (0,5 мкг/л - 1 мкг/л, фигура 20). В зоне «хэдспейс» контрольных штаммов, несущих вместо гена изопренсинтазы ген фитазы, изопрен не детектировался.
ПРИМЕР 7: Продуцирование изопрена в E.coli, экспрессирующей ген изопренсинтазы пуэрарии и гены idi, или dxs, или idi и dxs
I. Конструирование векторов, кодирующих ген изопренсинтазы пуэрарии и гены idi или dxs, или idi и dxs для продуцирования изопрена в E.coli
i) Конструирование pTrcKudzuKan
Ген bla pTrcKudzu (описанный в примере 1) заменяли геном, сообщающим резистентность к канамицину. Для удаления гена bla, плазмиду pTrcKudzu гидролизовали ферментом BspHI, обрабатывали щелочной фосфатазой креветок (SAP), подвергали термоинактивации при 65°С, а затем достраивали по концам с использованием фрагмента Кленова и dNTP. Крупный 5 т.п.н.-фрагмент выделяли из агарозного геля и лигировали с геном kanr, который подвергали ПЦР-амплификации из pCR-Blunt-II-TOPO с использованием праймеров MCM22 5'-GATCAAGCTTAACCGGAATTGCCAGCTG (SEQ ID NO:14) и MCM23 5'-GATCCGATCGTCAGAAGAACTCGTCAAGAAGGC (SEQ ID NO:15), гидролизовали ферментами HindIII и PvuI, и достраивали по концам. Трансформант, несущий плазмиду, сообщающую резистентность к канамицину (pTrcKudzuKan), отбирали на среде LA, содержащей 50 мкг/мл канамицина.
ii) Конструирование pTrcKudzu yIDI Kan
Плазмиду рTrcKudzuKan гидролизовали ферментом PstI, обрабатывали SAP, подвергали термоинактивации и очищали на геле. Затем плазмиду лигировали с ПЦР-продуктом, кодирующим ген idi от S. cerevisiae, содержащий синтетический RBS. Праймеры для ПЦР представляют собой NsiI-YIDI 1F 5'-CATCAATGCATCGCCCTTAGGAGGTAAAAAAAAATGAC (SEQ ID NO: 16) и PstI-YIDI 1R 5'-CCTTCTGCAGGACGCGTTGTTATAGC (SEQ ID NO: 17); а матрицей является геномная ДНК S. cerevisiae. ПЦР-продукт гидролизовали ферментами NsiI и PstI и подвергали гель-очистке, а затем лигировали. Смесь для лигирования переносили в химически компентеные клетки TOP10 и отбирали на среде LA, содержащей 50 мкг/мл канамицина. Несколько трансформантов выделяли и секвенировали, и полученную плазмиду обозначали pTrcKudzu-yΙDI(kan) (фигуры 34 и 35).
iii) Конструирование pTrcKudzu DXS Kan
Плазмиду рTrcKudzuKan гидролизовали ферментом PstI, обрабатывали SAP, подвергали термоинактивации и очищали на геле. Затем плазмиду лигировали с ПЦР-продуктом, кодирующим ген dxs E.coli, содержащий синтетический RBS. Праймеры для ПЦР представляют собой MCM13 5'-GATCATGCATTCGCCCTTAGGAGGTAAAAAAACATGAGTTTTGATATTGCCAAATACCCG (SEQ ID NO: 18) и MCM14 5'-CATGCTGCAGTTATGCCAGCCAGGCCTTGAT (SEQ ID NO: 19); а матрицей является геномная ДНК E.coli. ПЦР-продукт гидролизовали ферментами NsiI и PstI и подвергали гель-очистке, а затем лигировали. Смесь для трансформации переносили в клетки TOP10 и отбирали на среде LA, содержащей 50 мкг/мл канамицина. Несколько трансформантов выделяли и секвенировали, и полученную плазмиду обозначали pTrcKudzu-DXS(kan) (фигуры 36 и 37).
iv) Конструирование pTrcKudzu-yIDI-dxs (kan)
Плазмиду pTrcKudzu-yΙDI(kan) гидролизовали ферментом PstI, обрабатывали SAP, подвергали термоинактивации и очищали на геле. Затем плазмиду лигировали с ПЦР-продуктом, кодирующим ген dxs E.coli, содержащий синтетический RBS (праймеры представляют собой MCM13 5'-GATCATGCATTCGCCCTTAGGAGGTAAAAAAACATGAGTTTTGATATTG CCAAATACCCG (SEQ ID NO:18) и MCM14 5'-CATGCTGCAGTTATGCCAGCCAGGCC TTGAT (SEQ ID NO:19); а матрицей являются клетки ТОР10), где указанный продукт гидролизовали ферментами NsiI и PstI и подвергали гель-очистке. Конечную плазмиду обозначали pTrcKudzu-yIDI-dxs (kan) (фигуры 21 и 22).
v) Конструирование pCL PtrcKudzu
Фрагмент ДНК, содержащий промотор, структурный ген и терминатор, описанные выше в примере 1, гидролизовали из pTrcKudzu ферментом SspI и подвергали гель-очистке. Затем этот продукт лигировали с плазмидой pCL1920, которая была гидролизована ферментом PvuII, обработана SAP и подвергнута термоинактивации. Полученную смесь для лигирования переносили в клетки TOP10 и отбирали в среде LA, содержащей 50 мкг/мл спектиномицина. Несколько клонов выделяли и секвенировали, и отбирали два из этих клонов. Плазмиды pCL PtrcKudzu и pCL PtrcKudzu (A3) имели вставки в противоположных ориентациях (фигуры 38-41).
vi) Конструирование pCL PtrcKudzu yIDI
NsiI-PstI-гидролизованный и очищенный на геле ПЦР-ампликон IDI, полученный как описано выше в (ii), лигировали в плазмиду pCL PtrcKudzu, которая была гидролизована ферментом PstI, обработана SAP и подвергнута термоинактивации. Смесь для лигирования переносили в клетки TOP10 и отбирали в среде LA, содержащей 50 мкг/мл спектиномицина. Несколько клонов выделяли и секвенировали, и полученную плазмиду обозначали pCL PtrcKudzu yIDI (фигуры 42 и 43).
vii) Конструирование pCL PtrcKudzu DXS
NsiI-PstI-гидролизованный и очищенный на геле ПЦР-ампликон DXS, полученный как описано выше в (iii), лигировали в плазмиду pCL PtrcKudzu (А3), которая была гидролизована ферментом PstI, обработана SAP и подвергнута термоинактивации. Смесь для лигирования переносили в клетки TOP10 и отбирали в среде LA, содержащей 50 мкг/мл спектиномицина. Несколько клонов выделяли и секвенировали, и полученную плазмиду обозначали pCL PtrcKudzu DXS (фигуры 44 и 45).
II. Измерение уровня изопрена в зоне «хэдспейс» культур, экспрессирующих ген изопренсинтазы пуэрарии, idi и/или dxs с различным числом копий
Культуры E.coli BL21(λDE3), предварительно трансформированные плазмидами pTrcKudzu(kan) (A), pTrcKudzu-yIDI kan (B), pTrcKudzu-DXS kan (C), pTrcKudzu-yIDI-DXS kan (D), выращивали в LB-среде с канамицином, 50 мкг/мл. Культуры pCL PtrcKudzu (E), pCL PtrcKudzu, pCL PtrcKudzu-yIDI (F) и pCL PtrcKudzu-DXS (G) выращивали в LB с 50 мкг/мл спектиномицина. Культуры индуцировали 400 мкM IPTG на время 0 (OD600 примерно 0,5) и брали образцы для измерения уровня изопрена в зоне «хэдспейс» (см., пример 1). Результаты представлены на фигурах 23A-23G.
Плазмиду pTrcKudzu-yIDI-dxs (kan) вводили в штамм BL21 E.coli путем трансформации. Полученный штамм BL21/pTrc Kudzu IDI DXS культивировали в течение ночи в среде LB, содержащей канамицин (50 мкг/мл) при 20°С, и использовали для инокуляции шейкерных колб TM3 (13,6 г K2PO4, 13,6 г KH2PO4, 2,0 г MgSO4 · 7H2O), 2,0 г моногидрата лимонной кислоты, 0,3 г цитрата железа(3)-аммония, 3,2 г (NH4)2SO4, 0,2 г дрожжевого экстракта, 1,0 мл 1000 × модифицированного раствора следовых количеств металла, доведенного до pH 6,8 и до нужного объема путем добавления H2O, и стерилизованного на фильтре), содержащих 1% глюкозу. Колбы инкубировали при 30°С до OD600=0,8, а затем индуцировали 400 мкM IPTG. Образцы брали в различные периоды времени после индуцирования, и измеряли количество изопрена в зоне «хэдспейс», как описано в примере 1. Результаты представлены на фигуре 23H.
III. Продуцирование изопрена из биомассы в E.coli/pTrcKudzu yIDI DXS
Штамм BL21 pTrcKudzuIDIDXS тестировали на способность генерировать изопрен из биомассы трех типов: багассы, кукурузной соломы и пульпы мягкой древесины с использованием глюкозы в качестве контроля. Гидролизаты биомассы получали путем ферментативного гидролиза (Brown, L & Torget, R., 1996, NREL standard assay method Lap-009 "Enzymatic Saccharification of Lignocellulosic Biomass") и использовали при разведении на основе эквивалентов глюкозы. В этом примере, эквиваленты глюкозы равны 1% глюкозе. Моноколонию, взятую из планшета со свежетрансформированными клетками BL21 (DE3) pTrcKudzu yIDI DXS (kan), использовали для инокуляции 5 мл LB плюс канамицин (50 мкг/мл). Культуру инкубировали в течение ночи при 25°С со встряхиванием. На следующий день, ночную культуру разводили до OD600=0,05 в 25 мл TM3+0,2% YE+1% кормовой биомассы. Кормовая биомасса представляет собой кукурузную солому, багассу или пульпу мягкой древесины. В качестве позитивного контроля использовали глюкозу, а в качестве негативного контроля использовали образец, не содержащий глюкозу. Культуры инкубировали при 30°С со встряхиванием при 180 об/мин. Затем проводили мониторинг культуры на OD600, а когда OD600 достигала ~0,8, культуры анализировали на продуцирование изопрена в течение 1 и 3 часов, как описано в примере 1. Культуры не индуцировали. Все культуры, содержащие добавленную кормовую биомассу, продуцировали изопрен в количестве, эквивалентном количеству изопрена, содержащегося в позитивном контроле с глюкозой. Эксперименты, проведенные в дубликатах, представлены на фигуре 46.
IV. Продуцирование изопрена из инвертированного сахара в E.coli/pTrcKudzuIDIDXS
Моноколонию, взятую из планшета со свежетрансформированными клетками BL21 (λDE3)/pTrcKudzu yIDI DXS (kan), использовали для инокуляции 5 мл LB + канамицин (50 мкг/мл). Культуру инкубировали в течение ночи при 25°С со встряхиванием. На следующий день, ночную культуру разводили до OD600=0,05 в 25 мл TM3+0,2% YE+1% кормовой биомассы. Кормовая биомасса представляет собой глюкозу, инвертированную глюкозу или кукурузную солому. Кормовую биомассу, а именно, инвертированный сахар (инвертированный сахар Danisco) получали путем ферментативной обработки сахарного сиропа. Кукурузную солому AFEX получали, как описано ниже (часть V). Клетки культивировали при 30°С, и первый образец анализировали, когда культуры достигали OD600~0,8-1,0 (0 часов). Культуры анализировали на их рост по OD600 и на продуцирование изопрена, как описано в примере 1 через 0, 1 и 3 часа. Результаты представлены на фигуре 47.
V. Получение гидролизата из кукурузной соломы, предварительно обработанной AFEX
Кукурузную солому, предварительно обработанную AFEX, получали в Мичиганском институте биотехнологии. Предварительную обработку проводили при влажности 60%, загрузке аммиака 1:1, и при 90°С в течение 30 минут, с последующей сушкой воздухом. Содержание влаги в кукурузной соломе, предварительно обработанной AFEX, составляло 21,27%. Содержание глюкана и ксилана в кукурузной соломе, предварительно обработанной AFEX, составляло 31,7% и 19,1% (в расчете на сухую массу), соответственно. Процесс осахаривания осуществляли следующим образом: 20 г кукурузной соломы, предварительно обработанной AFEX, добавляли в 500 миллилитровую колбу с 5 мл 1 M натрийцитратного буфера, pH 4,8, 2,25 мл акселеразы (Accellerase) 1000, 0,1 мл Grindamyl H121 (продукт расщепления ксиланазой Aspergillus niger Danisco, используемый в хлебопекарной промышленности), и 72,65 мл дистиллированной воды DI. Колбу помещали в орбитальный шейкер и инкубировали при 50°С в течение 96 часов. Затем из шейкера брали один образец и анализировали с помощью ВЭЖХ. Гидролизат содержал 38,5 г/л глюкозы, 21,8 г/л ксилозы и 10,3 г/л олигомеров глюкозы и/или ксилозы.
VI. Влияние дрожжевого экстракта на продуцирование изопрена в клетках E.coli, культивируемых в периодической культуре с подпиткой
Ферментацию осуществляли в масштабе 14 литров, как описано выше, с использованием клеток E.coli, содержащих описанную выше плазмиду pTrcKudzu yIDI DXS. Дрожжевой экстракт (Bio Springer, Montreal, Quebec, Canada) подавали с экспоненциальной скоростью. В процессе 40-часовой ферментации, общее количество дрожжевого экстракта, подаваемое в ферментер, варьировалось в пределах 70-830 г. Оптическую плотность сбраживаемого бульона измеряли на длине волны 550 нм. Конечная оптическая плотность в ферментерах была пропорциональна количеству добавляемого дрожжевого экстракта (фигура 48A). Уровень изопрена в отходящем газе ферментера определяли, как описано выше. Титр изопрена увеличивался в процессе ферментации (фигура 48B). Количество продуцируемого изопрена было прямо пропорционально количеству подаваемого дрожжевого экстракта (фигура 48C).
VII. Продуцирование изопрена в 500-литровом ферментере при ферментации pTrcKudzu DXS yIDI
Для продуцирования изопрена использовали 500-литровый ферментер с клетками E.coli, содержащими нуклеиновые кислоты изопренсинтазы пуэрарии, IDI S. cerevisiae, и DXS E.coli (E.coli BL21 (λDE3) pTrc Kudzu dxs yidi). Уровни изопрена варьировались от 50 дo 300 мкг/л в течение 15 часов. Исходя из средних концентраций изопрена, средних значений для потока, проходящего через устройство, и уровня проходящего потока изопрена вычисляли количество собранного изопрена, которое составляло приблизительно 17 г.
VIII. Продуцирование изопрена в 500-литровом ферментере при ферментации клеток E.coli, культивированных в периодической культуре с подпиткой
Состав среды (на литр сбраживаемой среды):
7,5 г K2HPO4, 2 г МgSO4 · 7 Н2О, 2 г моногидрата лимонной кислоты, 0,3 г цитрата железа(3)-аммония, 0,5 г дрожжевого экстракта, 1 мл 1000 × модифицированного раствора следовых количеств металла. Все компоненты добавляли одновременно и растворяли в diH2O. Этот раствор автоклавировали. рН доводили до 7,0 добавлением газообразного аммония (NH3) и объем доводили до нужной величины. После стерилизации и коррекции рН добавляли 10 г глюкозы, 0,1 г тиамина · HCl и антибиотики.
1000 × модифицированный раствор следовых количеств металла:
40 г лимонной кислоты · Н2О; 30 г МnSO4 · Н2О; 10 г NaCl, 1 г FeSO4 · 7 Н2О; 1 г CoCl2 · 6 Н2О, 1 г ZnSO · 7 Н2О, 100 мг CuSO4 · 5 Н2О, 100 мг H3ВО3, 100 мг NaMoO4 · 2 Н2О. Каждый компонент сразу растворяли в diН2О, рН доводили до 3,0 добавлением HCl/NaOH, а затем объем доводили до нужной величины и стерилизовали на фильтре 0,22 микрон.
Ферментацию осуществляли в 500-литровом биореакторе с клетками E.coli, содержащими плазмиду pTrcKudzu yIDI DXS. Этот эксперимент проводили для мониторинга образования изопрена из глюкозы и дрожжевого экстракта при нужном рН ферментации, рН=7,0, и при температуре 30°С. Инокулят штамма E.coli, взятого из замороженного сосуда, приготавливали в среде, содержащей соевый пептон - дрожжевой экстракт - глюкозу. После культивирования инокулята до OD=0,15, измеренной на 550 нм, 20 мл этого инокулята использовали для инокуляции биореактора, содержащего 2,5 л среды, включающей соевый пептон - дрожжевой экстракт - глюкозу. Эти 2,5 л среды в биореакторе культивировали при 30°С до OD=1,0 и 2,0 л переносили в 500-литровый биореактор.
Дрожжевой экстракт (Bio Springer, Montreal, Quebec, Canada) и глюкозу подавали с экспоненциальной скоростью. В процессе 50-часовой ферментации, общее количество глюкозы и дрожжевого экстракта, подаваемое в биореактор, составляло 181,2 кг и 17,6 кг, соответственно. Оптическая плотность в биореакторе в течение всего периода времени представлена на фигуре 49А. Уровень изопрена в отходящем газе биореактора определяли, как описано выше. Титр изопрена увеличивался в процессе ферментации (фигура 49B). Общее количество продуцируемого изопрена в процессе 50-часовой ферментации составляло 55,1 г, а время продуцирования указано на фигуре 49C.
ПРИМЕР 8: Продуцирование изопрена в E.coli, экспрессирующей гены изопренсинтазы пуэрарии и рекомбинантные гены пути мевалоновой кислоты
I. Клонирование генов нижнего пути MVA
Стратегия клонирования генов нижнего пути мевалоновой кислоты описана ниже. Четыре гена пути биосинтеза мевалоновой кислоты, а именно, гены мевалонаткиназы (MVK), фосфомевалонаткиназы (PMK), дифосфомевалонатдекарбоксилазы (MVD), и изопентенилдифосфатизомеразы амплифицировали с помощью ПЦР из хромосомной ДНК S. cerevisiae и по отдельности клонировали в плазмиду pCR BluntII TOPO (Invitrogen). В некоторых случаях, ген idi амплифицировали из хромосомной ДНК E.coli. Праймеры конструировали так, чтобы консенсусная RBS (AGGAGGT (SEQ ID NO:80) или AAGGAGG (SEQ ID NO:81)) E.coli была встроена у 5'-конца, на 8 п.н. выше старт-кодона, а PstI-сайт был добавлен у 3'-конца. Затем гены один за другим клонировали в вектор pTrcHis2B до тех пор, пока не были собраны все гены этого пути.
Хромосомную ДНК от S. cerevisiae S288C получали из ATCC (ATCC 204508D). Ген MVK амплифицировали из хромосомы S. cerevisiae с использованием праймеров MVKF (5'-AGGAGGTAAAAAAACATGTCATTACCGTTCTTAACTTCTGC, SEQ ID NO:21) и MVK-Pst1-R (5'-ATGGCTGCAGGCCTATCGCAAATTAGCTTATGAAGTCCATGGTAAATTCGTG, SEQ ID NO:22) и полимеразы PfuTurbo в соответствии с инструкциями производителей. ПЦР-продукт нужного размера (1370 п.н.) идентифицировали путем электрофореза на 1,2% E-геле (Invitrogen) и клонировали в pZeroBLUNT TOPO. Полученную плазмиду обозначали pMVK1. Плазмиду pMVK1 гидролизовали рестриктирующими эндонуклеазами SacI и Taq1, а затем фрагмент очищали на геле и лигировали в плазмиду pTrcHis2B, гидролизованную ферментами SacI и BstBI. Полученную плазмиду обозначали pTrcMVK1.
Второй ген в пути биосинтеза мевалоновой кислоты, PMK, амплифицировали с помощью ПЦР с использованием нижеследующих праймеров: PstI-PMK1 R (5'-GAATTCGCCCTTCTGCAGCTACC, SEQ ID NO:23) и BsiHKA I-PMK1 F (5'-CGACTGGTGCACCCTTAAGGAGGAAAAAAACATGTCAG, SEQ ID NO:24). ПЦР-реакцию осуществляли с использованием полимеразы Pfu Turbo (Stratagene) в соответствии с инструкциями производителей. Продукт нужного размера (1387 п.н.) гидролизовали ферментами PstI и BsiHKI и лигировали в плазмиду pTrcMVK1, гидролизованную ферментом PstI. Полученную плазмиду обозначали pTrcKK. Гены MVD и idi клонировали тем же самым способом. ПЦР осуществляли с использованием пар праймеров PstI-MVD 1R (5'-GTGCTGGAATTCGCCCTTCTGCAGC, SEQ ID NO:25) и NsiI-MVD 1F (5'-GTAGATGCATGCAGAATTCGCCCTTAAGGAGG, SEQ ID NO:26) для амплификации гена MVD и PstI-YIDI 1R (5'-CCTTCTGCAGGACGCGTTGTTATAGC, SEQ ID NO:27) и NsiI-YIDI 1F (5'-CATCAATGCATCGCCCTTAGGAGGTAAAAAAAAATGAC, SEQ ID NO:28) для амплификации гена yIDI. В некоторых случаях использовали ген IPP-изомеразы, idi от E.coli. Для амплификации гена idi из хромосомной ДНК E.coli использовали набор нижеследующих праймеров: PstI-CIDI 1R (5'-GTGTGATGGATATCTGCAGAATTCG, SEQ ID NO:29) и NsiI-CIDI 1F (5'-CATCAATGCATCGCCCTTAGGAGGTAAAAAAACATG, SEQ ID NO:30). В качестве матричной ДНК служила хромосомная ДНК, выделенная стандартными методами из E.coli FM5 (WO 96/35796 и WO 2004/033646, которые во всей своей полноте вводятся в настоящее описание посредством ссылки, а в частности, в описание выделения нуклеиновых кислот). Конечные плазмиды обозначали pKKDIy для конструкции, содержащей дрожжевой ген idi, или pKKDIc для конструкции, содержащей ген idi E.coli. Эти плазмиды переносили в клетки-хозяева BL21 E.coli для последующего анализа. В некоторых случаях, ген изопренсинтазы пуэрарии клонировали в pKKDIy с получением плазмиды pKKDIyIS.
Ген нижнего пути MVA также клонировали в плазмиду pTrc, содержащую маркер резистентности к антибиотику канамицину. Плазмиду pTrcKKDIy гидролизовали рестриктирующими эндонуклеазами ApaI и PstI, и 5930 п.н. - фрагмент разделяли на 1,2% агарозном E-геле и очищали с помощью набора для гель-очистки Qiagen в соответствии с инструкциями производителей. Плазмиду pTrcKudzuKan, описанную в примере 7, гидролизовали рестриктирующими эндонуклеазами ApaI и PstI, и вектор, содержащий 3338 п.н. - фрагмент, очищали из 1,2% E-геля с помощью набора для гель-очистки Qiagen. 3338 п.н. - фрагмент вектора и 5930 п.н. - фрагмент нижнего пути MVA лигировали с использованием набора для лигирования Roche Quick. Смесь для лигирования переносили в клетки ТОР10 E.coli, и трансформанты культивировали при 37°С в течение ночи, а затем отбирали на среде LA, содержащей канамицин (50 мкг/мл). Трансформанты подтверждали путем гидролиза рестриктирующими ферментами, и один из них замораживали для использования в качестве маточного раствора. Эту плазмиду обозначали pTrcKanKKDIy.
II. Клонирование гена изопренсинтазы пуэрарии в pTrcKanKKDIy
Ген изопренсинтазы пуэрарии амплифицировали с помощью ПЦР из плазмиды pTrcKudzu, полученной, как описано в примере 1, с использованием праймеров MCM50 5'-GATCATGCATTCGCCCTTAGGAGGTAAAA AAACATGTGTGCGACCTCTTCTCAATTTACT (SEQ ID NO:31) и MCM53 5'-CGGTCGACGGATCCCTGCAGTTAGACATACATCAGCTG (SEQ ID NO:32). Полученный ПЦР-фрагмент клонировали в pCR2.1 и переносили в TOP10 E.coli. Этот фрагмент содержал последовательность, кодирующую изопренсинтазу пуэрарии и вышерасположенную область, содержащую RBS E.coli. Трансформанты инкубировали в течение ночи при 37°С и отбирали на среде LA, содержащей карбенициллин (50 мкг/мл). Нужную вставку фрагмента подтверждали путем секвенирования, и этот штамм обозначали MCM93.
Плазмиду штамма MCM93 гидролизовали рестриктирующими эндонуклеазами NsiI и PstI для выделения 1724 п.н. - вставки, содержащей RBS и изопренсинтазу пуэрарии. 1724 п.н. - фрагмент разделяли на 1,2% агарозном E-геле и очищали с помощью набора для гель-очистки Qiagen в соответствии с инструкциями производителей. Плазмиду pTrcKanKKDIy гидролизовали рестриктирующей эндонуклеазой PstI, обрабатывали SAP в течение 30 минут при 37°С и очищали с помощью набора для ПЦР-очистки Qiagen. Плазмиду и фрагмент ДНК, кодирующий изопренсинтазу пуэрарии, лигировали с использованием набора для лигирования Roche Quick. Смесь для лигирования переносили в клетки ТОР10 E.coli, и трансформанты культивировали при 37°С в течение ночи, а затем отбирали на среде LA, содержащей канамицин (50 мкг/мл). Нужный трансформант подтверждали путем гидролиза рестриктирующими ферментами, и плазмиду обозначали pTrcKKDyIkISKan (фигуры 24 и 25). Эту плазмиду переносили в клетки BL21(λDE3) (Invitrogen).
III. Продуцирование изопрена из мевалоната в клетках E.coli, экспрессирующих рекомбинантный ген нижнего мелованатного пути и изопренсинтазы пуэрарии
Штамм BL21/pTrcKKDyIkISKan культивировали в среде MOPS (Neidhardt et al., (1974) J. Bacteriology 119:736-747), рН которой был доведен до pH 7,1, и в которую были добавлены 0,5% глюкоза и 0,5% мевалоновая кислота. Контрольную культуру также получали в идентичных условиях, но без добавления 0,5% мевалоновой кислоты. В качестве исходной культуры брали ночную посевную культуру, в которую добавляли 1% инокулят, и которую индуцировали 500 мкM IPTG, когда эта культура достигала OD600=0,3-0,5. Культуры выращивали при 30°С со встряхиванием при 250 об/мин. Продуцирование изопрена анализировали через 3 часа после индуцирования с помощью анализа зоны «хэдспейс», как описано в примере 1. Максимальное продуцирование изопрена составляло 6,67×10-4 моль/лбульона/OD6oo/час, где лбульона представляет собой объем бульона и включает объем клеточной среды и объем клеток. Контрольная культура, в которую не добавляли мевалоновую кислоту, не продуцировала какого-либо детектируемого количества изопрена.
IV. Клонирование генов верхнего пути MVA
Гены верхнего пути биосинтеза мевалоната, а именно два гена, кодирующие три ферментативные активности, клонировали из Enterococcus faecalis. Ген mvaE кодирует белок, обладающий ферментативными активностями ацетил-CoA-ацетилтрансферазы и 3-гидрокси-3-метилглутарил-CoA (HMG-CoA)-редуктазы, первого и третьего белков в этом пути, а ген mvaS кодирует второй фермент в этом пути, HMG-CoA-синтазу. Ген mvaE амплифицировали из геномной ДНК E. faecalis (ATCC 700802D-5), содержащей сайт связывания с рибосомой E.coli и расположенный перед ним спейсер, с использованием нижеследующих праймеров:
CF 07-60 (+) старт-кодон mvaE с RBS + старт-кодон ATG SacI
5'-GAGACATGAGCTCAGGAGGTAAAAAAACATGAAAACAGTAGTTATTATTG (SEQ ID NO:34)
CF 07-62 (-) лигирование mvaE с mvaS с RBS между ними
5'-TTTATCAATCCCAATTGTCATGTTTTTTTACCTCCTTTATTGTTTTCTTAAATC (SEQ ID NO:35)
Ген mvaS амплифицировали из геномной ДНК E. faecalis (ATCC 700802D-5), содержащей RBS и расположенный перед ним спейсер E.coli, с использованием нижеследующих праймеров:
CF 07-61 (+) лигирование mvaE с mvaS с RBS между ними
5'-GATTTAAGAAAACAATAAAGGAGGTAAAAAAACATGACAATTGGGATTGATAAA (SEQ ID NO:36)
CF 07-102 (-) конец гена mvaS-BglII
5'-GACATGACATAGATCTTTAGTTTCGATAAGAACGAACGGT (SEQ ID NO:37)
ПЦР-фрагменты лигировали вместе с помощью ПЦР с использованием нижеследующих праймеров:
CF 07-60 (+) старт-кодон mvaE с RBS + старт-кодон ATG SacI
5'-GAGACATGAGCTCAGGAGGTAAAAAAACATGAAAACAGTAGTTATTATTG (SEQ ID NO:34)
CF 07-102 (-)конец гена mvaS-BglII
5'-GACATGACATAGATCTTTAGTTTCGATAAGAACGAACGGT (SEQ ID NO:37)
Гибридный ПЦР-фрагмент очищали с помощью набора Qiagen и гидролизовали рестриктирующими ферментами SacI и BglII. Этот гидролизованный фрагмент ДНК подвергали гель-очистке с помощью набора Qiagen и лигировали в коммерчески доступный вектор pTrcHis2A, который был гидролизован ферментами SacI и BglII и подвергнут гель-очистке.
Смесь для лигирования переносили в клетки E. coli Top 10 и колонии отбирали на планшетах с LA+50 мкг/мл карбенициллина. Было отобрано всего шесть колоний, которые затем культивировали в течение ночи в LB+50 мкг/мл карбенициллина, и плазмиды выделяли с использованием набора Qiagen. Плазмиды гидролизовали ферментами SacI и BglII для анализа на вставки, и одну нужную плазмиду секвенировали с использованием нижеследующих праймеров:
CF 07-58 (+) старт-кодон гена mvaE
5'-ATGAAAACAGTAGTTATTATTGATGC (SEQ ID NO:38)
CF 07-59 (-) конец гена mvaE
5'-ATGTTATTGTTTTCTTAAATCATTTAAAATAGC (SEQ ID NO:39)
CF 07-82 (+)старт-кодон гена mvaS
5'-ATGACAATTGGGATTGATAAAATTAG (SEQ ID NO:40)
CF 07-83 (-) конец гена mvaS
5'-TTAGTTTCGATAAGAACGAACGGT (SEQ ID NO:41)
CF 07-86 (+) последовательность в mvaE
5'-GAAATAGCCCCATTAGAAGTATC (SEQ ID NO:42)
CF 07-87 (+) последовательность в mvaE
5'-TTGCCAATCATATGATTGAAAATC (SEQ ID NO:43)
CF 07-88 (+) последовательность в mvaE
5'-GCTATGCTTCATTAGATCCTTATCG (SEQ ID NO:44)
CF 07-89 (+)последовательность в mvaS
5'-GAAACCTACATCCAATCTTTTGCCC (SEQ ID NO:45)
Плазмиду, обозначенную pTrcHis2AUpperPathway#1, подтверждали путем секвенирования и переносили в коммерчески доступный штамм BL21 E.coli. Отбор проводили на среде LA+50 мкг/мл карбенициллина. В результате отбирали два трансформанта и культивировали на среде LB+50 мкг/мл карбенициллина до тех пор, пока они не достигали OD600=1,5. Оба штамма замораживали в сосуде при -80°С в присутствии глицерина. Эти штаммы обозначали CF 449 для pTrcHis2AUpperPathway#1 в BL21, изолят #1, и CF 450 для pTrcHis2AUpperPathway#1 в BL21, изолят #2. Анализ показал, что оба клона были идентичны.
V. Клонирование гена верхнего пути MVA в pCL 1920
Плазмиду pTrcHis2AUpperPathway гидролизовали рестриктирующей эндонуклеазой SspI с получением фрагмента, содержащего pTrc-mvaE-mvaS-(His-метка)-терминатор. В этом фрагменте, his-метка не транслировалась. Этот затупленный по концам 4,5 т.п.н. - фрагмент очищали из 1,2% E-геля с использованием набора для гель-очистки Qiagen. Дефосфорилированный и затупленный по концам 4,2 т.п.н. - фрагмент от pCL1920 получали путем гидролиза вектора рестриктирующей эндонуклеазой PvuII, обработки SAP и очистки из 1,2% E-геля с помощью набора для гель-очистки Qiagen. Два фрагмента лигировали с использованием набора для лигирования Roche Quick и переносили в химически компетентные клетки TOP10. Трансформанты отбирали на среде LA, содержащей спектиномицин (50 мкг/мл). Нужную колонию идентифицировали путем скрининга на присутствие вставки с помощью ПЦР. Плазмиду обозначали pCL PtrcUpperPathway (фигуры 26 и 27A-27D).
VI. Штаммы, экспрессирующие гены комбинированных верхних и нижних путей мевалоновой кислоты
Для получения штамма, содержащего все гены пути мевалоновой кислоты и изопренсинтазы пуэрарии, плазмиды pTrcKKDyIkISkan и pCLpTrcUpperPathway переносили в компетентные клетки BL21(λDE3) (Invitrogen), и трансформанты отбирали на среде LA, содержащей канамицин (50 мкг/мл) и спектиномицин (50 мкг/мл). Трансформанты подтверждали с использованием плазмидных препаратов для гарантии того, что обе эти плазмиды сохранялись в хозяине. Этот штамм обозначали MCM127.
VII. Продуцирование мевалоновой кислоты из глюкозы в E.coli/pUpperpathway
Моноколонии BL21/pTrcHis2A-mvаE/mvаS или FM5/p pTrcHis2A-mvaE/mvaS инокулировали в среду LB + карбенициллин (100 мкг/мл) и культивировали в течение ночи при 37°С со встряхиванием при 200 об/мин. Эти культуры разводили в 50 мл среды в 250 мл-колбах с перегородкой до OD600=0,1. Эта среда представляет собой TM3+1 или 2% глюкоза + карбенициллин (100 мкг/мл) или TM3+1% глюкоза + гидролизованное соевое масло + карбенициллин (100 мкг/мл) или TM3 + биомасса (полученная багасса, кукурузная солома или просо). Культуры выращивали при 30°С со встряхиванием при 200 об/мин приблизительно в течение 2-3 часов до тех пор, пока OD600 не достигала 0,4. На этой стадии, экспрессию конструкции mvaEmvaS индуцировали добавлением IPTG (400 мкM). Культуры инкубировали еще 20 или 40 часов с образцами, взятыми через 2-6 часов после индуцирования, а затем через 24, 36 и 48 часов, если это необходимо. Взятие образцов осуществляли путем забора 1 мл культуры, измерения OD600, осаждения клеток в микроцентрифуге, удаления супернатанта и анализа на мевалоновую кислоту.
Клетки E.coli, содержащие нуклеиновые кислоты, кодирующие полипептиды AA-CoA-тиолазы, HMG-CoA-синтазы и HMG-CoA-редуктазы Enterococcus faecalis, при их ферментации в объеме 14 литров в клеточной среде TM3 и 2% глюкозе, используемой в качестве клеточной среды, продуцировали 22 грамма мевалоновой кислоты. В шейкерных колбах, эти клетки продуцировали 2-4 грамма мевалоновой кислоты на литр среды LB с 1% глюкозой, используемой в качестве клеточной культуральной среды. Продуцирование мевалоновой кислоты в этих штаммах указывало на то, что путь MVA является функциональным в E.coli.
VIII. Продуцирование изопрена из E.coli BL21, содержащих ген верхнего и нижнего пути MVA и ген изопренсинтазы пуэрарии
Нижеследующие штаммы получали путем трансформации различных комбинаций плазмид, содержащих ген верхнего и нижнего пути MVA и ген изопренсинтазы пуэрарии, как описано выше, и плазмид, содержащих гены idi, dxs и dxr и изопренсинтазы, как описано в примере 7. Используемыми клетками-хозяевами были химически компетентные BL21(λDE3), а трансформанты были получены стандартными методами. Трансформанты отбирали на L-агаре, содержащем канамицин (50 мкг/мл) или канамицин + спектиномицин (оба в концентрациях 50 мкг/мл). Планшеты культивировали при 37°С. Полученные штаммы имели следующие обозначения:
Штаммы, культивируемые на канамицине + спектиномицине (50 мкг/мл каждого)
MCM127 - pCL Upper MVA + pTrcKKDyIkIS (kan) в BL21(λDE3)
MCM131 - pCL1920 + pTrcKKDyIkIS (kan) в BL21(λDE3)
MCM125 - pCL Upper MVA + pTrcHis2B (kan) в BL21(λDE3)
Штаммы, культивируемые на канамицине (50 мкг/мл)
MCM64 - pTrcKudzu yIDI DXS (kan) в BL21(λDE3)
MCM50 - pTrcKudzu (kan) в BL21(λDE3)
MCM123 - pTrcKudzu yIDI DXS DXR (kan) в BL21(λDE3)
Вышеуказанные штаммы, взятые из замороженных маточных растворов, высевали штрихом на среде LA + соответствующий антибиотик, и культивировали в течение ночи при 37°С. Одну колонию от каждого планшета использовали для инокуляции шейкерных колб (25 мл LB + соответствующий антибиотик). Колбы инкубировали при 22°С в течение ночи со встряхиванием при 200 об/мин. На следующее утро, колбы переносили в инкубатор при 37°С и культивировали еще 4,5 часа со встряхиванием при 200 об/мин. 25 мл культур центрифугировали для осаждения клеток, и клетки ресуспендировали в 5 мл LB + соответствующий антибиотик. Затем культуры разводили в 25 мл LB+1% глюкоза + соответствующий антибиотик до OD600=0,1. Для каждого штамма приготавливали две колбы, одну из которых индуцировали IPTG (800 мкM), а другую не индуцировали. Культуры инкубировали при 37°С со встряхиванием при 250 об/мин. Одну серию этих культур индуцировали через 1,50 часа (сразу после взятия образца на момент времени 1). При каждом взятии образца измеряли OD600 и определяли количество изопрена, как описано в примере 1. Результаты представлены в таблице 3. Количество продуцируемого изопрена представляет собой количество при максимальном продуцировании для конкретного штамма.
следовые количества: пик присутствует, но не поддается интегрированию.
IX. Анализ мевалоновой кислоты
Мевалонолактон (1,0 г, 7,7 ммоль) (CAS#503-48-0) закупали у Sigma-Aldrich (WI, USA) в виде сиропа, который растворяли в воде (7,7 мл) и обрабатывали гидроксидом калия (7,7 ммоль) для получения калиевой соли мевалоновой кислоты. Превращение в мевалоновую кислоту подтверждали с помощью 1H-ЯМР-анализа. Образцы для ВЭЖХ-анализа получали путем центрифугирования при 14000 об/мин в течение 5 минут для удаления клеток, а затем 300 мкл-аликвоту супернатанта добавляли к 900 мкл H2O. После этого добавляли перхлорную кислоту (36 мкл 70% раствора), а затем смесь перемешивали и охлаждали на льду в течение 5 минут. После этого образцы снова центрифугировали (14000 об/мин в течение 5 минут) и супернатант переносили в ВЭЖХ-колонку. Стандарты мевалоновой кислоты (20, 10, 5, 1 и 0,5 г/л) получали аналогичным образом. Анализ мевалоновой кислоты (с объемом впрыска 20 мкл) проводили с помощью ВЭЖХ на колонке BioRad Aminex 87-H+ (300 мм × 7,0 мм) с элюированием 5 мM серной кислотой при скорости 0,6 мл/мин и с определением показателя преломления (RI). В этих условиях, мевалоновая кислота элюировалась в форме лактона за 18,5 минут.
X. Продуцирование изопрена из клеток BL21 E.coli, содержащих ген верхнего пути MVA + ген изопренсинтазы пуэрарии
Для продуцирования изопрена из клеток в периодической культуре с подпиткой, клетки E.coli, экспрессирующие полипептиды пути мевалоновой кислоты и изопренсинтазу пуэрарии, подвергали ферментации в масштабе 15 литров. Этот эксперимент продемонстрировал, что культивирование клеток в условиях с ограниченным содержанием глюкозы приводило к продуцированию 2,2 г/л изопрена.
Состав среды (на литр сбраживаемой среды):
Среду получали с использованием нижеследующих компонентов на литр сбраживаемой среды: 7,5 г K2HPO4, 2 г МgSO4 · 7 Н2О, 2 г моногидрата лимонной кислоты, 0,3 г цитрата железа(3)-аммония, 0,5 г дрожжевого экстракта, 1 мл 1000 × модифицированного раствора следовых количеств металла. Все компоненты добавляли одновременно и растворяли в diH2O. Этот раствор автоклавировали. рН доводили до 7,0 добавлением гидроксида аммония (30%) и объем доводили до нужной величины. После стерилизации и коррекции рН добавляли 10 г глюкозы, 0,1 г тиамина · HCl и антибиотики.
1000 × модифицированный раствор следовых количеств металла:
1000 × модифицированный раствор следовых количеств металла получали с использованием нижеследующих компонентов: 40 г лимонной кислоты · Н2О; 30 г МnSO4 · Н2О; 10 г NaCl, 1 г FeSO4 · 7 Н2О; 1 г CoCl2 · 6 Н2О, 1 г ZnSO · 7 Н2О, 100 мг CuSO4 · 5 Н2О, 100 мг H3ВО3, 100 мг NaMoO4 · 2 Н2О. Каждый компонент сразу растворяли в diН2О, рН доводили до 3,0 добавлением HCl/NaOH, а затем объем доводили до нужной величины и стерилизовали на фильтре 0,22 микрон.
Ферментацию осуществляли в 15-литровом биореакторе с клетками E.coli BL21 (DE3), содержащими плазмиды pCL PtrcUpperPathway (фигура 26) и pTrcKKDyIkIS. Этот эксперимент проводили для мониторинга образования изопрена из глюкозы при нужном рН ферментации, рН=7,0, и при температуре 30°С. Инокулят штамма E.coli, взятый из замороженного сосуда, высевали штрихом на агаровый планшет с бульоном LB (в присутствии антибиотиков) и инкубировали при 37°С. Одну колонию инокулировали в среду, содержащую соевый пептон - дрожжевой экстракт - глюкозу. После культивирования инокулята до OD=1,0, измеренной на 550 нм, 500 мл этого инокулята использовали для инокуляции 5-литрового биореактора.
Глюкозу подавали с экспоненциальной скоростью до тех пор, пока клетки не достигали стационарной фазы. После этого, скорость подачи глюкозы снижали так, чтобы это удовлетворяло метаболическим требованиям. Общее количество глюкозы, подаваемое в биореактор в процессе 54-часовой ферментации, составляло 3,7 кг. Индуцирование достигалось путем добавления изопропил-бета-D-1-тиогалактопиранозида (IPTG). Когда оптическая плотность на 550 нм (OD550) достигала значения 10, концентрацию IPTG доводили до 25 мкМ. Когда OD550 достигала 190, концентрацию IPTG доводили до 50 мкМ. После 38-часовой ферментации, концентрацию IPTG повышали до 100 мкM. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 54. Уровень изопрена в отходящем газе биореактора определяли, как описано в настоящей заявке. В процессе ферментации, титр изопрена увеличивался до конечной величины 2,2 г/л (фиг. 55). Общее количество изопрена, продуцируемое в течение 54-часовой ферментации, составляло 15,9 г, а время продуцирования указано на фиг. 56.
XI. Ферментация изопрена из клеток E.coli, экспрессирующих гены пути мевалоновой кислоты и культивируемых в периодической культуре с подпиткой в масштабе 15 литров
Для продуцирования изопрена из клеток в периодической культуре с подпиткой, клетки E.coli, экспрессирующие полипептиды пути мевалоновой кислоты и изопренсинтазу пуэрарии, подвергали ферментации в масштабе 15 литров. Этот эксперимент продемонстрировал, что культивирование клеток в условиях с ограниченным содержанием глюкозы приводило к продуцированию 3,0 г/л изопрена.
Состав среды (на литр сбраживаемой среды):
Среду получали с использованием нижеследующих компонентов на литр сбраживаемой среды: 7,5 г K2HPO4, 2 г МgSO4 · 7 Н2О, 2 г моногидрата лимонной кислоты, 0,3 г цитрата железа(3)-аммония, 0,5 г дрожжевого экстракта, 1 мл 1000 × модифицированного раствора следовых количеств металла. Все компоненты добавляли одновременно и растворяли в diH2O. Этот раствор автоклавировали. рН доводили до 7,0 добавлением гидроксида аммония (30%) и объем доводили до нужной величины. После стерилизации и коррекции рН добавляли 10 г глюкозы, 0,1 г тиамина · HCl и антибиотики.
1000 × модифицированный раствор следовых количеств металла:
1000 × модифицированный раствор следовых количеств металла получали с использованием нижеследующих компонентов: 40 г лимонной кислоты · Н2О; 30 г МnSO4 · Н2О; 10 г NaCl, 1 г FeSO4 · 7 Н2О; 1 г CoCl2 · 6 Н2О, 1 г ZnSO · 7 Н2О, 100 мг CuSO4 · 5 Н2О, 100 мг H3ВО3, 100 мг NaMoO4 · 2 Н2О. Каждый компонент сразу растворяли в diН2О, рН доводили до 3,0 добавлением HCl/NaOH, а затем объем доводили до нужной величины и стерилизовали на фильтре 0,22 микрон.
Ферментацию осуществляли в 15-литровом биореакторе с клетками E.coli BL21 (DE3), содержащими плазмиды pCL PtrcUpperMVA и pTrcKKDyIkIS. Этот эксперимент проводили для мониторинга образования изопрена из глюкозы при нужном рН ферментации, рН=7,0, и при температуре 30°С. Инокулят штамма E.coli, взятый из замороженного сосуда, высевали штрихом на агаровый планшет с бульоном LB (в присутствии антибиотиков) и инкубировали при 37°С. Одну колонию инокулировали в среду, содержащую триптон - дрожжевой экстракт. После культивирования инокулята до OD=1,0, измеренной на 550 нм, 500 мл этого инокулята использовали для инокуляции 5-литрового биореактора.
Глюкозу подавали с экспоненциальной скоростью до тех пор, пока клетки не достигали стационарной фазы. После этого, скорость подачи глюкозы снижали так, чтобы это удовлетворяло метаболическим требованиям. Общее количество глюкозы, подаваемое в биореактор в процессе 59-часовой ферментации, составляло 2,2 кг. Индуцирование достигалось путем добавления IPTG. Когда оптическая плотность на 550 нм (OD550) достигала значения 10, концентрацию IPTG доводили до 25 мкМ. Когда OD550 достигала 190, концентрацию IPTG доводили до 50 мкМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 93. Уровень изопрена в отходящем газе биореактора определяли, как описано в настоящей заявке. В процессе ферментации, титр изопрена увеличивался до конечной величины 3,0 г/л (фиг. 94). Общее количество изопрена, продуцируемое в течение 59-часовой ферментации, составляло 22,8 г, а время продуцирования указано на фиг. 95. Молярный выход утилизованного углерода, который превращался в изопрен в процессе ферментации, составлял 2,2%. Выход изопрена в процентах по массе глюкозы составлял 1,0%.
XII. Ферментация изопрена из клеток E.coli, экспрессирующих гены пути мевалоновой кислоты и культивируемых в периодической культуре с подпиткой в масштабе 15 литров
Для продуцирования изопрена из клеток в периодической культуре с подпиткой, клетки E.coli, экспрессирующие полипептиды пути мевалоновой кислоты, изопренсинтазу Pueraria lobata, и изопренсинтазу пуэрарии волосистой, подвергали ферментации в масштабе 15 литров. Этот эксперимент продемонстрировал, что культивирование клеток в условиях с ограниченным содержанием глюкозы приводило к продуцированию 3,3 г/л изопрена.
i) Конструирование плазмиды pCLPtrcUpperPathwayHGS2
Ген, кодирующий изопренсинтазу Pueraria lobata, подвергали ПЦР-амплификации с использованием праймеров NsiI-RBS-HGS F (CTTGATGCATCCTGCATTCGCCCTTAGGAGG, SEQ ID NO:88) и pTrcR (CCAGGCAAATTCTGTTTTATCAG, SEQ ID NO:89), и pTrcKKDyIkIS в качестве матрицы. Полученный таким образом ПЦР-продукт гидролизовали рестриктирующими ферментами NsiI и PstI и подвергали гель-очистке. Плазмиду pCL PtrcUpperPathway гидролизовали рестриктирующим ферментом PstI и дефосфорилировали с использованием щелочной фосфатазы rAPid (Roche) в соответствии с инструкциями производителей.
Эти ДНК-фрагменты лигировали вместе, и реакционную смесь для лигирования переносили в химически компетентные клетки E.coli Top 10 (Invitrogen), высевали на L-агар, содержащий спектиномицин (50 мкг/мл), и инкубировали в течение ночи при 37°С. Плазмидную ДНК получали из 6 клонов с использованием набора Qiaquick Spin Mini-prep. Плазмидную ДНК гидролизовали рестриктирующими ферментами EcoRV и MluI для идентификации клона, в котором вставка имеет правильную ориентацию (то есть ген ориентирован в таком же направлении, как и промотор pTrc).
Полученную нужную плазмиду обозначали pCLPtrcUpperPathwayHGS2. Эту плазмиду анализировали с помощью анализа в зоне «хэдспейс», описанного в настоящей заявке, и было обнаружено, что она продуцирует изопрен в E.coli Top 10, что подтверждает функциональность данного гена. Эту плазмиду переносили в клетки BL21(LDE3), содержащие pTrcKKDyIkIS, и получали штамм BL21/pCLPtrcUpperPathwayHGS2-pTrcKKDyIkIS. Этот штамм, по сравнению со штаммом BL21/pCL PtrcUpperMVA и штаммом pTrc KKDyIkIS, имел дополнительную копию изопренсинтазы (пример 8, часть XI). Указанный штамм, по сравнению со штаммом BL21/pCL PtrcUpperMVA и штаммом pTrc KKDyIkIS, используемыми в примере 8, части XI, также экспрессировал HMGS на более высоком уровне и обладал повышенной HMGS-активностью.
ii) Ферментация изопрена из клеток E.coli, экспрессирующих pCLPtrcUpperPathwayHGS2-pTrcKKDyIkIS и культивируемых в периодической культуре с подпиткой в масштабе 15 литров
Состав среды (на литр сбраживаемой среды):
Среду получали с использованием нижеследующих компонентов на литр сбраживаемой среды: 7,5 г K2HPO4, 2 г МgSO4 · 7 Н2О, 2 г моногидрата лимонной кислоты, 0,3 г цитрата железа(3)-аммония, 0,5 г дрожжевого экстракта, и 1 мл 1000 × модифицированного раствора следовых количеств металла. Все компоненты добавляли одновременно и растворяли в diH2O. Этот раствор автоклавировали. рН доводили до 7,0 добавлением гидроксида аммония (30%) и объем доводили до нужной величины. После стерилизации и коррекции рН добавляли 10 г глюкозы, 0,1 г тиамина · HCl и антибиотики.
1000 × модифицированный раствор следовых количеств металла:
1000 × модифицированный раствор следовых количеств металла получали с использованием нижеследующих компонентов: 40 г лимонной кислоты · Н2О; 30 г МnSO4 · Н2О; 10 г NaCl, 1 г FeSO4 · 7 Н2О; 1 г CoCl2 · 6 Н2О, 1 г ZnSO · 7 Н2О, 100 мг CuSO4 · 5 Н2О, 100 мг H3ВО3, 100 мг NaMoO4 · 2 Н2О. Каждый компонент сразу растворяли в diН2О, рН доводили до 3,0 добавлением HCl/NaOH, а затем объем доводили до нужной величины и стерилизовали на фильтре 0,22 микрон.
Ферментацию осуществляли в 15-литровом биореакторе с клетками E.coli BL21 (DE3), содержащими плазмиды pCLPtrcUpperPathwayHGS2 и pTrcKKDyIkIS. Этот эксперимент проводили для мониторинга образования изопрена из глюкозы при нужном рН ферментации, рН=7,0, и при температуре 30°С. Инокулят штамма E.coli, взятый из замороженного сосуда, высевали штрихом на агаровый планшет с бульоном LB (в присутствии антибиотиков) и инкубировали при 37°С. Одну колонию инокулировали в среду, содержащую триптон - дрожжевой экстракт. После культивирования инокулята до OD=1,0, измеренной на 550 нм, 500 мл этого инокулята использовали для инокуляции 5-литрового биореактора.
Глюкозу подавали с экспоненциальной скоростью до тех пор, пока клетки не достигали стационарной фазы. После этого, скорость подачи глюкозы снижали так, чтобы это удовлетворяло метаболическим требованиям. Общее количество глюкозы, подаваемое в биореактор в процессе 58-часовой ферментации, составляло 2,1 кг. Индуцирование достигалось путем добавления IPTG. Когда оптическая плотность на 550 нм (OD550) достигала значения 9, концентрацию IPTG доводили до 25 мкМ. Когда OD550 достигала 170, концентрацию IPTG доводили до 50 мкМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 104. Уровень изопрена в отходящем газе биореактора определяли, как описано в настоящей заявке. В процессе ферментации, титр изопрена увеличивался до конечной величины 3,3 г/л (фиг. 105). Общее количество изопрена, продуцируемое в течение 58-часовой ферментации, составляло 24,5 г, а время продуцирования указано на фиг. 106. Молярный выход утилизованного углерода, который превращался в изопрен в процессе ферментации, составлял 2,5%. Выход изопрена в процентах по массе глюкозы составлял 1,2%. Анализ показал, что активность изопренсинтазы увеличивалась приблизительно в 3-4 раза по сравнению с активностью клеток BL21, экспрессирующих плазмиды CL PtrcUpperMVA и pTrc KKDyIkIS (данные не приводятся).
XIII. Интеграция генов нижнего мевалонатного пути в хромосому E.coli
Синтетический оперон, содержащий ген мевалонаткиназы, мевалонатфосфаткиназы, мевалонатпирофосфатдекарбоксилазы и IPP-изомеразы, интегрировали в хромосому E.coli. При желании, экспрессия может быть модифицирована путем интеграции различных промоторов у 5'-конца оперона.
В таблице 9 перечислены праймеры, используемые в этом эксперименте.
i) Конструирование нужного вектора
attTn7-сайт отбирали для интеграции. Области гомологии, расположенные выше (attTn7 up) (праймеры MCM78 и MCM79) и ниже (attTn7 down) (праймеры MCM88 и MCM89) амплифицировали с помощью ПЦР из клеток MG1655. 50 мкл реакционной смеси, содержащей 1 мкл 10 мкM праймеров, 3 мкл ddH2O, 45 мкл смеси для ПЦР Invitrogen Platinum PCR Supermix High Fidelity, и колонию MG1655, полученную путем соскоба, денатурировали в течение 2 минут при 94°C, за 25 циклов (2 минуты при 94°C, 30 секунд при 50°C, и 1 минута при 68°C), с последующим удлинением в течение 7 минут при 72°C, и охлаждением до 4°C. Полученную ДНК клонировали в pCR2.1 (Invitrogen) в соответствии с инструкциями производителей, в результате чего получали плазмиды MCM278 (attTn7 up) и MCM252 (attTn7 down). 832 п.н.-ApaI-PvuI-фрагмент, который был гидролизован и очищен на геле из MCM252, клонировали в ApaI-PvuI-гидролизованную и очищенную на геле плазмиду pR6K, в результате чего получали плазмиду MCM276. 825 п.н.-PstI-NotI-фрагмент, который был гидролизован и очищен на геле из MCM278, клонировали в PstI-NotI-гидролизованную и очищенную на геле плазмиду MCM276, и получали плазмиду MCM281.
ii) Клонирование гена и промотора нижнего пути
Гены MVK-PMK-MVD-IDI амплифицировали из pTrcKKDyIkIS с использованием праймеров MCM 104 и MCM 105 с применением ПЦР-системы Roche Expand Long в соответствии с инструкциями производителей. Этот продукт гидролизовали ферментами NotI и ApaI и клонировали в плазмиду MCM281, которая была гидролизована ферментами NotI и ApaI и очищена на геле. Праймеры MCM120 и MCM127 были использованы для амплификации кластера CMR из матричной ДНК GeneBridges FRT-gb2-Cm-FRT с использованием полимеразы Stratagene Pfu Ultra II. ПЦР-программа включала денатурацию при 95°C, 4 минуты, 5 циклов: 95°C, 20 секунд, 55°C, 20 секунд, 72°C, 2 минуты, 25 циклов: 95°C, 20 секунд, 58°C, 20 секунд, 72°C, 2 минуты, 72°C, 10 минут, а затем четыре 50 мкл ПЦР-смеси, содержащей 1 мкл ~10 нг/мкл матрицы, 1 мкл каждого праймера, 1,25 мкл 10 мМ dNTP, 5 мкл 10x буфера, 1 мкл фермента и 39,75 мкл ddH2О, охлаждали до 4°С. Реакционные смеси объединяли, очищали на колонке для ПЦР-очистки Qiagen и использовали для электропорации промытых водой клеток Pirl, содержащих плазмиду MCM296. Электропорацию осуществляли в 2 мM-кюветах при 2,5 В и 200 Ом. Реакционные смеси для электропорации регенерировали в LB в течение 3 часов при 30°C. Трансформант MCM330 отбирали на LA с CMP5, Kan50 (фигуры 107 и 108A-108C).
iii) Интеграция в хромосому E.coli
Минипрепарат ДНК (набор Qiaquick Spin) от MCM330 гидролизовали ферментом SnaBI и использовали для электропорации клеток BL21(DE3) (Novagen) или MG1655, содержащих плазмиду pRedET Carb GeneBridges. Клетки культивировали при 30°С до OD ~1, а затем индуцировали 0,4% L-арабинозой при 37°C в течение 1,5 часа. Эти клетки три раза промывали ddH2O при 4°С, а затем подвергали электропорации с использованием 2 мкл ДНК. Интегранты отбирали на L-агаре в присутствии хлорамфеникола (5 мкг/мл), а затем подтверждали, что они не растут на L-агаре + канамицин (50 мкг/мл). Интегрант BL21 MCM331 и интегрант MG 1655 MCM333 замораживали.
iv) Конструирование плазмиды pET24D-Kudzu, кодирующей изопренсинтазу пуэрарии
Ген изопренсинтазы пуэрарии субклонировали в вектор pET24d (Novagen) из вектора pCR2.1 (Invitrogen). В частности, ген изопренсинтазы пуэрарии амплифицировали из матричной ДНК pTrcKudzu с использованием праймеров: MCM50 5'-GATCATGCAT TCGCCCTTAG GAGGTAAAAA AACATGTGTG CGACCTCTTC TCAATTTACT (SEQ ID NO:99) и MCM53 5'-CGGTCGACGG ATCCCTGCAG TTAGACATAC ATCAGCTG (SEQ ID NO: 100). ПЦР-реакции осуществляли с использованием ДНК-полимеразы Taq (Invitrogen), и полученный ПЦР-продукт клонировали в клонирующий вектор pCR2.1-TOPO TA (Invitrogen), а затем переносили в химически компетентные клетки E.coli Top 10 (Invitrogen). Трансформанты высевали на L-агар, содержащий карбенициллин (50 мкг/мл), и инкубировали в течение ночи при 37°С. Пять миллилитров культур бульона Лурия, содержащего 50 мкг/мл карбенициллина, инокулировали одиночными трансформантами и культивировали в течение ночи при 37°С. Пять колоний скринировали на нужную вставку путем секвенирования плазмидной ДНК, выделенной из 1 мл жидкой культуры (бульона Лурия), и очищали с помощью набора QIAprep Spin Mini-prep (Qiagen). Полученная плазмида, обозначенная MCM93, содержала последовательность, кодирующую изопренсинтазу пуэрарии в остове pCR2.1.
Последовательность, кодирующую изопренсинтазу пуэрарии, удаляли путем гидролиза рестриктирующими эндонуклеазами PciI и BamH1 (Roche) и подвергали гель-очистке с использованием набора для экстракции геля QIAquick (Qiagen). ДНК вектора pET24d гидролизовали ферментами NcoI и BamH1 (Roche), обрабатывали щелочной фосфатазой креветок (Roche), и очищали с использованием набора QIAprep Spin Mini-prep (Qiagen). Фрагмент гена изопренсинтазы пуэрарии лигировали с NcoI/BamH1-гидролизованной плазмидой pET24d с использованием набора для быстрого лигирования ДНК (Roche) в отношении фрагмент:вектор = 5:1 в общем объеме 20 мкл. Часть смеси для лигирования (5 мкл) переносили в химически компетентные клетки E.coli Top 10 и высевали на L-агар, содержащий канамицин (50 мкг/мл). Нужный трансформант подтверждали путем секвенирования и переносили в химически компетентные клетки BL21(λDE3)pLysS (Novagen). Моноколонию отбирали после культивирования в течение ночи при 37°C на L-агаре, содержащем канамицин (50 мкг/мл). Карта полученной плазмиды, обозначенной pET24D-Kudzu, представлена на фигуре 109. Последовательность pET24D-Kudzu (SEQ ID NO:101) представлена на фигурах 110А и 110B. Изопренсинтазную активность подтверждали с помощью анализа в зоне «хэдспейс».
v) Штаммы-продуценты
Штаммы MCM331 и MCM333 котрансформировали плазмидами pCLPtrcupperpathway и либо pTrcKudzu, либо pETKudzu, в результате чего получали штаммы, представленные в таблице 10.
vi) Ферментация изопрена из клеток E.coli, экспрессирующих гены пути мевалоновой кислоты и культивируемых в периодической культуре с подпиткой в масштабе 15 литров
Состав среды (на литр сбраживаемой среды):
Среду получали с использованием нижеследующих компонентов на литр сбраживаемой среды: 7,5 г K2HPO4, 2 г МgSO4 · 7 Н2О, 2 г моногидрата лимонной кислоты, 0,3 г цитрата железа(3)-аммония, 0,5 г дрожжевого экстракта и 1 мл 1000 × модифицированного раствора следовых количеств металла. Все компоненты добавляли одновременно и растворяли в diH2O. Этот раствор автоклавировали. рН доводили до 7,0 добавлением гидроксида аммония (30%) и объем доводили до нужной величины. После стерилизации и коррекции рН добавляли 10 г глюкозы, 0,1 г тиамина · HCl и антибиотики.
1000 × модифицированный раствор следовых количеств металла:
1000 × модифицированный раствор следовых количеств металла получали с использованием нижеследующих компонентов: 40 г лимонной кислоты · Н2О; 30 г МnSO4 · Н2О; 10 г NaCl, 1 г FeSO4 · 7 Н2О; 1 г CoCl2 · 6 Н2О, 1 г ZnSO · 7 Н2О, 100 мг CuSO4 · 5 Н2О, 100 мг H3ВО3, 100 мг NaMoO4 · 2 Н2О. Каждый компонент сразу растворяли в diН2О, рН доводили до 3,0 добавлением HCl/NaOH, а затем объем доводили до нужной величины и стерилизовали на фильтре 0,22 микрон.
Ферментацию осуществляли в 15-литровом биореакторе с клетками E.coli BL21 (DE3), содержащими интегрированную плазмиду нижнего пути MVA gi1.2, описанную выше, и плазмиды pCLPtrcUpperMVA и pTrcKudzu. Этот эксперимент проводили для мониторинга образования изопрена из глюкозы при нужном рН ферментации, рН=7,0, и при температуре 30°С. Инокулят штамма E.coli, взятый из замороженного сосуда, высевали штрихом на агаровый планшет с бульоном LB (в присутствии антибиотиков) и инкубировали при 37°С. Одну колонию инокулировали в среду, содержащую триптон - дрожжевой экстракт. После культивирования инокулята до OD=1,0, измеренной на 550 нм, 500 мл этого инокулята использовали для инокуляции 5-литрового биореактора.
Глюкозу подавали с экспоненциальной скоростью до тех пор, пока клетки не достигали стационарной фазы. После этого, скорость подачи глюкозы снижали так, чтобы это удовлетворяло метаболическим требованиям. Общее количество глюкозы, подаваемое в биореактор в процессе 57-часовой ферментации, составляло 3,9 кг. Индуцирование достигалось путем добавления IPTG. Когда скорость выделения диоксида углерода достигала 100 ммоль/л/час, концентрацию IPTG доводили до 100 мкМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 111А. Уровень изопрена в отходящем газе биореактора определяли, как описано в настоящей заявке. В процессе ферментации, титр изопрена увеличивался до конечной величины 1,6 г/л (фиг. 111В). Удельная продуктивность изопрена в процессе ферментации указана на фиг. 111С и достигала максимума при 1,2 мг/OD/час. Общее количество изопрена, продуцируемое в течение 57-часовой ферментации, составляло 16,2 г. Молярный выход утилизованного углерода, который превращался в изопрен в процессе ферментации, составлял 0,9%. Выход изопрена в процентах по массе глюкозы составлял 0,4%.
XIV. Продуцирование изопрена из клеток E.coli BL21, содержащих изопренсинтазу пуэрарии, с использованием глицерина в качестве источника углерода
Продуцирование изопрена из клеток E.coli, экспрессирующих изопренсинтазу пуэрарии, осуществляли путем ферментации в периодической культуре с подпиткой глицерином в масштабе 15 литров. Этот эксперимент показал, что культивирование клеток в присутствии глицерина (без глюкозы) приводило к продуцированию 2,2 мг/л изопрена.
Состав среды (на литр сбраживаемой среды):
Среду получали с использованием нижеследующих компонентов на литр сбраживаемой среды: 7,5 г K2HPO4, 2 г МgSO4 · 7 Н2О, 2 г моногидрата лимонной кислоты, 0,3 г цитрата железа(3)-аммония, и 1 мл 1000 × модифицированного раствора следовых количеств металла. Все компоненты добавляли одновременно и растворяли в diH2O. Этот раствор автоклавировали. рН доводили до 7,0 добавлением гидроксида аммония (30%) и объем доводили до нужной величины. После стерилизации и коррекции рН добавляли 5,1 г глицерина, 0,1 г тиамина · HCl и антибиотики.
1000 × модифицированный раствор следовых количеств металла:
Данную среду получали с использованием нижеследующих компонентов на литр сбраживаемой среды: 40 г лимонной кислоты · Н2О; 30 г МnSO4 · Н2О; 10 г NaCl, 1 г FeSO4 · 7 Н2О; 1 г CoCl2 · 6 Н2О, 1 г ZnSO · 7 Н2О, 100 мг CuSO4 · 5 Н2О, 100 мг H3ВО3, 100 мг NaMoO4 · 2 Н2О. Каждый компонент сразу растворяли в diН2О, рН доводили до 3,0 добавлением HCl/NaOH, а затем объем доводили до нужной величины и стерилизовали на фильтре 0,22 микрон.
Ферментацию осуществляли в 15-литровом биореакторе с клетками E.coli BL21 (DE3), содержащими плазмиду pTrcKudzu. Этот эксперимент проводили для мониторинга образования изопрена из глицерина при нужном рН ферментации, рН=7,0, и при температуре 35°С. Инокулят штамма E.coli, взятый из замороженного сосуда, высевали штрихом на агаровый планшет с бульоном LА (в присутствии антибиотиков) и инкубировали при 37°С. Одну колонию инокулировали в среду, содержащую соевый пептон - дрожжевой экстракт-глюкозу и культивировали при 35°С. После культивирования инокулята до OD=1,0, измеренной на 550 нм, 600 мл этого инокулята использовали для инокуляции 7,5-литрового биореактора.
Глицерин подавали с экспоненциальной скоростью до тех пор, пока оптическая плотность на 550 нм (OD550) не достигала 153. Общее количество глицерина, подаваемое в биореактор в процессе 36-часовой ферментации, составляло 1,7 кг. В инокуляте не присутствовала глюкоза, и глюкозу также не добавляли в биореактор. Индуцирование достигалось путем добавления IPTG. Когда OD550 достигала 50, концентрацию IPTG доводили до 20 мкМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 57. Уровень изопрена в отходящем газе биореактора определяли, как описано в настоящей заявке. В процессе ферментации, титр изопрена увеличивался до конечной величины 2,2 мг/л (фиг. 58). Общее количество изопрена, продуцируемое в течение 54-часовой ферментации, составляло 20,9 мг, а время продуцирование указано на фиг. 59.
XV. Ферментация изопрена из клеток E.coli, экспрессирующих гены пути мевалоновой кислоты и культивируемых в периодической культуре с подпиткой в масштабе 15 литров с использованием инвертированного сахара в качестве источника углерода
Продуцирование изопрена из клеток E.coli, экспрессирующих полипептиды пути мевалоновой кислоты и изопренсинтазу пуэрарии, осуществляли путем ферментации в периодической культуре с подпиткой инвертированным сахаром в масштабе 15 литров. Этот эксперимент показал, что культивирование клеток в присутствии инвертированного сахара приводило к продуцированию 2,4 г/л изопрена.
Состав среды (на литр сбраживаемой среды):
Среду получали с использованием нижеследующих компонентов на литр сбраживаемой среды: 7,5 г K2HPO4, 2 г МgSO4 · 7 Н2О, 2 г моногидрата лимонной кислоты, 0,3 г цитрата железа(3)-аммония, 0,5 г дрожжевого экстракта и 1 мл 1000 × модифицированного раствора следовых количеств металла. Все компоненты добавляли одновременно и растворяли в diH2O. Этот раствор автоклавировали. рН доводили до 7,0 добавлением гидроксида аммония (30%) и объем доводили до нужной величины. После стерилизации и коррекции рН добавляли 10 г инвертированного сахара, 0,1 г тиамина · HCl и антибиотики.
1000 × модифицированный раствор следовых количеств металла:
1000 × модифицированный раствор следовых количеств металла получали с использованием нижеследующих компонентов: 40 г лимонной кислоты · Н2О; 30 г МnSO4 · Н2О; 10 г NaCl, 1 г FeSO4 · 7 Н2О; 1 г CoCl2 · 6 Н2О, 1 г ZnSO · 7 Н2О, 100 мг CuSO4 · 5 Н2О, 100 мг H3ВО3, 100 мг NaMoO4 · 2 Н2О. Каждый компонент сразу растворяли в diН2О, рН доводили до 3,0 добавлением HCl/NaOH, а затем объем доводили до нужной величины и стерилизовали на фильтре 0,22 микрон.
Ферментацию осуществляли в 15-литровом биореакторе с клетками E.coli BL21 (DE3), содержащими плазмиды pCLPtrcUpperMVA и pTrc KKDyIkIS. Этот эксперимент проводили для мониторинга образования изопрена из инвертированного сахара при нужном рН ферментации, рН=7,0, и при температуре 30°С. Инокулят штамма E.coli, взятый из замороженного сосуда, высевали штрихом на агаровый планшет с бульоном LB (в присутствии антибиотиков) и инкубировали при 37°С. Одну колонию инокулировали в среду, содержащую триптон - дрожжевой экстракт. После культивирования инокулята до OD=1,0, измеренной на 550 нм, 500 мл этого инокулята использовали для инокуляции 5-литрового биореактора.
Инвертированный сахар подавали с экспоненциальной скоростью до тех пор, пока клетки не достигали стационарной фазы. После этого, скорость подачи глюкозы снижали так, чтобы это удовлетворяло метаболическим требованиям. Общее количество инвертированного сахара, подаваемое в биореактор в процессе 44-часовой ферментации, составляло 2,4 кг. Индуцирование достигалось путем добавления IPTG. Когда оптическая плотность на 550 мМ (OD550) достигала 9, концентрацию IPTG доводили до 25 мкМ. Когда OD550 достигала 200, концентрацию IPTG доводили до 50 мкМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 96. Уровень изопрена в отходящем газе биореактора определяли, как описано в настоящей заявке. В процессе ферментации, титр изопрена увеличивался до конечной величины 2,4 г/л (фиг. 97). Общее количество изопрена, продуцируемое в течение 44-часовой ферментации, составляло 18,4 г, а время продуцирования указано на фиг. 98. Молярный выход утилизованного углерода, который превращался в изопрен в процессе ферментации, составлял 1,7%. Выход изопрена в процентах по массе глюкозы составлял 0,8%.
ПРИМЕР 9. Конструирование генов верхнего и нижнего пути MVA для интеграции в Bacillus subtilis
I. Конструирование генов верхнего и нижнего пути MVA в Bacillus subtilis
Ген верхнего пути от Enterococcus faecalis интегрировали в B. subtilis под контролем промотора aprE. Верхний путь состоит из двух генов: гена mvaE, который кодирует AACT и HMGR, и гена mvaS, который кодирует HMGS. Эти два гена лигировали вместе со стоп-кодоном, расположенным между ними, и сайтом RBS, расположенным перед mvaS, и эти гены находились под контролем промотора aprE. Терминатор расположен за геном mvaE. Маркер резистентности к хлорамфениколу клонировали за геном mvaE, и эту конструкцию интегрировали в виде локуса aprE посредством двойного кроссинговера с использованием фланкирующих областей гомологии.
Четыре ДНК-фрагмента амплифицировали с помощью ПЦР так, чтобы они содержали выступающие концы, позволяющие осуществлять лигирование этих фрагментов с помощью ПЦР-реакции. ПЦР-амплификацию осуществляли с использованием полимеразы Herculase в соответствии с инструкциями производителей.
1. PaprE
CF 07-134 (+) Старт-кодон промотора aprE-PstI
5'- GACATCTGCAGCTCCATTTTCTTCTGC (SEQ ID NO: 82)
CF 07-94 (-) Лигирование PaprE с mvaE
5'- CAATAATAACTACTGTTTTCACTCTTTACCCTCTCCTTTTAA (SEQ ID NO: 83)
Матрица: хромосомная ДНК Bacillus subtilis
2. mvaE
CF 07-93 (+) Лигирование mvaE с промотором aprE (старт-кодон GTG)
5'-TTAAAAGGAGAGGGTAAAGAGTGAAAACAGTAGTTATTATTG (SEQ ID NO: 84)
CF 07-62 (-) Лигирование mvaE с mvaS с RBS, расположенным между ними
5'-TTTATCAATCCCAATTGTCATGTTTTTTTACCTCCTTTATTGTTTTCTTAAATC (SEQ ID NO:35)
Матрица: хромосомная ДНК Enterococcus faecalis (из ATCC)
3. mvaS
CF 07-61 (+) Лигирование mvaE с mvaS с RBS, расположенным между ними
5'-GATTTAAGAAAACAATAAAGGAGGTAAAAAAACATGACAATTGGGATTGATAAA (SEQ ID NO:36)
CF 07-124 (-) Лигирование конца mvaS с терминатором
5'-CGGGGCCAAGGCCGGTTTTTTTTAGTTTCGATAAGAACGAACGGT (SEQ ID NO:85)
Матрица: хромосомная ДНК Enterococcus faecalis
4. Терминатор щелочной сериновой протеазы B. amyliquefaciens
CF 07-123 (+) Лигирование конца mvaS с терминатором
5'-ACCGTTCGTTCTTATCGAAACTAAAAAAAACCGGCCTTGGCCCCG (SEQ ID NO:86)
CF 07-46 (-) Конец терминатора B. amyliquefaciens - BamHI
5'-GACATGACGGATCCGATTACGAATGCCGTCTC (SEQ ID NO:63)
Матрица: хромосомная ДНК Bacillus amyliquefaciens
Реакция ПЦР-лигирования
5. Лигирование mvaE с mvaS
CF 07-93 (+) Лигирование mvaE с промтором aprE (старт-кодон GTG)
5'-TTAAAAGGAGAGGGTAAAGAGTGAAAACAGTAGTTATTATTG (SEQ ID NO: 84)
CF 07-124 (-) Лигирование конца mvaS с терминатором
5'-CGGGGCCAAGGCCGGTTTTTTTTAGTTTCGATAAGAACGAACGGT (SEQ ID NO:85)
Матрица: #2 и 3 от вышеуказанных конструкций
6. Лигирование mvaE-mvaS с промотором aprE
CF 07-134 (+) Старт-кодон промотора aprE-PstI
5'- GACATCTGCAGCTCCATTTTCTTCTGC (SEQ ID NO: 82)
CF 07-124 (-) Лигирование конца mvaS с терминатором
5'-CGGGGCCAAGGCCGGTTTTTTTTAGTTTCGATAAGAACGAACGGT (SEQ ID NO:85)
Матрица: #1 и 4 от вышеуказанных конструкций
7. Лигирование PaprE-mvaE-mvaS с терминатором
CF 07-134 (+) Старт-кодон промотора aprE-PstI
5'-GACATCTGCAGCTCCATTTTCTTCTGC (SEQ ID NO: 82)
CF 07-46 (-) Конец терминатора B. amyliquefaciens-BamHI
5'-GACATGACGGATCCGATTACGAATGCCGTCTC (SEQ ID NO:63)
Матрица: #4 и #6
Продукт гидролизовали рестриктирующими эндонуклеазами PstI/BamHI и лигировали с плазмидой pJM102 (Perego, M. 1993. Integrational vectors for genetic manipulation in Bacillus subtilis, p. 615-624. In A. L. Sonenshein, J. A. Hoch, и R. Losick (ed.), Bacillus subtilis and other gram-positive bacteria: biochemistry, physiology, and molecular genetics. American Society for Microbiology, Washington, D.C.), которую гидролизовали ферментами PstI/BamHI. Смесь для лигирования переносили в химически компетентные клетки E.coli TOP 10, и трансформанты отбирали на среде LA, содержащей карбенициллин (50 мкг/мл). Нужную плазмиду идентифицировали путем секвенирования и обозначали pJMUpperpathway2 (фигуры 50 и 51). Очищенную плазмидную ДНК переносили в клетки Bacillus subtilis aprEnprE Pxyl-comK, и трансформанты отбирали на L-агаре, содержащем хлорамфеникол (5 мкг/мл). Нужную колонию отбирали, а затем высевали на L-агар, содержащий 10, 15 и 25 мкг/мл хлорамфеникола, для амплификации ряда копий кластера, содержащего ген верхнего пути.
Полученный штамм тестировали на продуцирование мевалоновой кислоты путем его культивирования в среде LB, содержащей 1% глюкозу и 1% (?). Культуры анализировали с помощью ГХ на продуцирование мевалоновой кислоты.
Этот штамм затем использовали в качестве хозяина для интеграции нижнего пути мевалоновой кислоты.
Для секвенирования различных вышеуказанных конструкций были использованы следующие праймеры:
Секвенирующие праймеры:
CF 07-134 (+) Старт-кодон промотора aprE-PstI
5'-GACATCTGCAGCTCCATTTTCTTCTGC (SEQ ID NO: 82)
CF 07-58 (+) Старт-кодон гена mvaE
5'-ATGAAAACAGTAGTTATTATTGATGC (SEQ ID NO:38)
CF 07-59 (-) Конец гена mvaE
5'-ATGTTATTGTTTTCTTAAATCATTTAAAATAGC (SEQ ID NO:39)
CF 07-82 (+)Старт-кодон гена mvaS
5'-ATGACAATTGGGATTGATAAAATTAG (SEQ ID NO:40)
CF 07-83 (-) Конец гена mvaS
5'-TTAGTTTCGATAAGAACGAACGGT (SEQ ID NO:41)
CF 07-86 (+) Последовательность в mvaE
5'-GAAATAGCCCCATTAGAAGTATC (SEQ ID NO:42)
CF 07-87 (+) Последовательность в mvaE
5'-TTGCCAATCATATGATTGAAAATC (SEQ ID NO:43)
CF 07-88 (+) Последовательность в mvaE
5'-GCTATGCTTCATTAGATCCTTATCG (SEQ ID NO:44)
CF 07-89 (+) Последовательность mvaS
5'-GAAACCTACATCCAATCTTTTGCCC (SEQ ID NO:45)
Трансформанты отбирали на среде LA, содержащей хлорамфеникол в концентрации 5 мкг/мл. Одну колонию подтверждали на правильную интеграцию путем секвенирования и высевали на среду LA, содержащую возрастающие концентрации хлорамфеникола, и оставляли на несколько дней до конечного уровня 25 мкг/мл. В результате этого осуществлялась амплификация кластера, содержащего представляющие интерес гены. Полученный штамм обозначали CF 455: pJMupperpathway#1 X Bacillus subtilis aprEnprE Pxy1 comK (амплифицированный для роста на среде LA, содержащей 25 мкг/мл хлорамфеникола).
II. Конструирование гена нижнего пути MVA в Bacillus subtilis
Нижний путь MVA, состоящий из генов MVK1, pmk, mpd и idi, объединяли в кластер, состоящий из фланкирующих областей ДНК, происходящих от области nprE хромосомы B. subtilis (сайт интеграции), из промотора aprE и маркера резистентности к спектиномицину (см. фигуры 28 и 29). Этот кластер был синтезирован фирмой DNA2.0 и интегрирован в хромосому B. subtilis, содержащую ген верхнего пути MVA, интегрированный в локусе aprE. Ген изопренсинтазы пуэрарии экспрессировался из реплицирующейся плазмиды, описанной в примере 4, и был перенесен в штамм с интегированными генами верхнего и нижнего путей.
ПРИМЕР 10: Репрезентативные композиции изопрена и способы их получения
I. Общий анализ состава отходящего газа, образующегося при ферментации и содержащего изопрен
Ферментацию в масштабе 14 литров осуществляли с использованием рекомбинантного штамма BL21 (DE3) E.coli, содержащего две плазмиды (pCL upperMev; pTrcKKDyIkIS), кодирующие весь мевалонатный путь биосинтеза изопреноидного предшественника, дрожжевую изопренилпирофосфат-изомеразу и изопренсинтазу пуэрарии. Отходящий газ, образующийся при ферментации в 14-литровом реакторе, собирали в 20 мл-сосуды в зоне «хэдспейс», приблизительно в период максимальной продуцируемости изопрена (время ферментации 27,9 часов, «EFT») и анализировали с помощью ГХ/МС в зоне «хэдспейс» на летучие компоненты.
Анализ в зоне «хэдспейс» осуществляли с использованием ГХ/МС-системы Agilent 6890, снабженной ГХ/МС-колонкой Agilent HP-5MS (30 м × 250 мкм; толщина пленки 0,25 мкм). Для взятия образцов 500 мкл-аликвот из 20 мл-сосудов в зоне «хэдспейс» использовали автоматический инжектор combiPAL. В ГХ/МС-методе, в качестве газа-носителя используется гелий при скорости потока 1 мл/мин. Отверстие для впрыска поддерживали при 250°С, а отношение деления потока составляло 50:1. Температуру печи в первые две минуты поддерживали при 37°С, а затем ее повышали до 237°С со скоростью 25°С/мин, а общее время проведения метода составляло 10 минут. Селективный масс-спектрометрический детектор Agilent 5793N сканировал m/z в пределах от m/z 29 дo m/z 300. Предел детектирования данной системы составлял приблизительно 0,1 мкг/лгаза или приблизительно 0,1 м.д. При желании может быть использовано более чувствительное оборудование с меньшим пределом детектирования.
Отходящий газ состоял из 99,925% (об/об) постоянных газов (N2, CO2 и O2), приблизительно 0,075% изопрена (2-метил-1,3-бутадиена) (~750 м.д. по объему, 2100 мкг/л) и небольших количеств (<50 м.д. по объему) этанола, ацетона и двух прениловых С5-спиртов. Количество водяного пара не определяли, но по оценкам, оно должно быть равно равновесному давлению пара при 0°С. Состав фракции органических летучих веществ определяли путем интегрирования кривой пиков для получения площади под этими пиками на ГХ/МС-хроматограмме (фиг. 86A и 86B), и этот состав указан в таблице 6. Калибровочные кривые для этаноловых и ацетоновых стандартов позволяют преобразовывать ГХ-площадь в концентрацию газовой фазы, выраженную в мкг/л, в соответствии со стандартными методами.
Таблица 6. Состав смеси летучих органических компонентов отходящего газа, образующегося при ферментации.
Отходящий газ анализировали после 27,9-часовой ферментации с использованием штамма BL21 (DE3) E.coli, экспрессирующего гетерологичный мевалонатный путь, изопренилпирофосфат-изомеразу дрожжей и изопренсинтазу пуэрарии.
II. Определение следовых количеств летучих органических соединений (VOC), продуцируемых вместе с изопреном в процессе ферментации рекомбинантного штамма E.coli
Ферментацию в масштабе 14 литров осуществляли с использованием рекомбинантного штамма BL21 (DE3) E.coli, содержащего две плазмиды (pCL upperMev; pTrcKKDyIkIS), кодирующие весь мевалонатный путь биосинтеза изопреноидного предшественника, дрожжевую изопренилпирофосфат-изомеразу и изопренсинтазу пуэрарии.
Отходящий газ, образующийся в процессе ферментации, пропускали через охлажденные сосуды в зоне «хэдспейс» для определения концентрации и для идентификации следовых количеств летучих органических компонентов. Затем брали образцы отходящего газа при ферментации, проходящего со скоростью 1 л/минуту в течение 10 минут через 20 мл-сосуд в зоне «хэдспейс», упакованный кварцевой ватой (2 г), и охлаждали до -78°С на сухом льду. Затем сосуд закрывали новой крышкой и анализировали с помощью ГХ/МС-анализа в зоне «хэдспейс» на захват летучих органических компонентов (VОC) в условиях, описанных в примере 10, часть I. Соотношения соединений, указанных на фиг. 87A-87D, представляют собой комбинацию общего уровня отходящего газа при ферментации, относительного давления пара при -78°C, и ответа, детектируемого масс-спектрометром. Так, например, низкий уровень изопрена по отношению к окисленным летучим веществам (например, ацетону и этанолу) является показателем высокой летучести этого вещества, которое не аккумулируется в сосуде в зоне «хэдспейс» при -78°С.
Присутствие многих из этих соединений является уникальным для композиций изопрена, происходящих от биологических источников. Результаты представлены на фиг. 87A-87D и систематизированы в таблицах 7A и 7B.
2 % площади означает площадь пика, выраженную в % по отношению к общей площади пика для всех соединений.
3 Отношение (%) означает площадь пика, выраженную в % по отношению к площади пика для 2-метил-1,3-бутадиена.
2 % площади означает площадь пика, выраженную в % по отношению к общей площади пика для всех соединений.
3 Отношение (%) означает площадь пика, выраженную в % по отношению к площади пика для 2-метил-1,3-бутадиена.
III. Отсутствие изомеров C5-углеводородов в изопрене, образующемся в процессе ферментации
Крио-захват изопрена, присутствующего в отходящем газе при ферментации, осуществляли с использованием 2 мл-сосудов в зоне «хэдспейс», охлажденных в жидком азоте. Отходящий газ (1 л/мин) сначала пропускали через 20 мл-сосуд, содержащий таблетки гидроксида натрия для минимизации аккумуляции льда и твердого CO2 в 2 мл-сосуде (-196°C). Через этот сосуд пропускали приблизительно 10 л отходящего газа, после чего его нагревали до -78°С путем вентиляции сосуда, а затем его снова герметично закрывали другой крышкой и анализировали с помощью ГХ/МС.
Анализ зоны «хэдспейс» с помощью ГХ/МС осуществляли на ГХ/МС-системе Agilent 6890 с использованием газонепроницаемого шприца емкостью 100 мкл в режиме «хэдспейс». Разделение аналитов проводили на ГХ/МС-колонке (30 м × 250 мкм; пленка толщиной 1,40 мкм) Zebron ZB-624. Автоматический ГХ-инжектор подсоединяли к газонепроницаемому шприцу емкостью 100 мкл, а длину иглы корректировали на впрыск 50 мкл образца зоны «хэдспейс», взятого из 2-миллилитрового ГХ-сосуда. В этом ГХ/МС-методе, в качестве газа-носителя использовали гелий со скоростью потока 1 мл/мин. Отверстие для впрыска поддерживали при 200°С, а отношение деления потока составляло 20:1. Температуру печи в течение первых 5 минут проведения анализа поддерживали при 37°С. Селективный масс-спектрометрический детектор Agilent 5793N работал в режиме мониторинга одного иона (SIM) при m/z 55, 66, 67 и 70. В этих условиях элюирование изопрена происходило за 2,966 минут (фиг. 88B). Этим методом также анализировали стандартный изопрен, полученный из нефти (Sigma-Aldrich), и было обнаружено, что он содержит дополнительные C5-углеводородные изомеры, которые сразу элюировались до или после главного пика и были количественно оценены исходя из скорректированной ГХ-площади (фиг. 88A).
В отдельном эксперименте был проведен анализ стандартной смеси C5-углеводородов для того, чтобы определить, является ли детектируемый ответ одинаковым для каждого соединения. Указанными соединениями являются 2-метил-1-бутен, 2-метил-1,3-бутадиен, (E)-2-пентен, (Z)-2-пентен и (E)-1,3-пентадиен. В этом случае, анализ проводили на колонке Agilent DB-Petro (100 м × 0,25 мм, толщина пленки 0,50 мкм) при 50°С в течение 15 минут. В этом ГХ/МС-методе, в качестве газа-носителя использовали гелий со скоростью потока 1 мл/мин. Отверстие для впрыска поддерживали при 200°С, а отношение деления потока составляло 50:1. Селективный масс-спектрометрический детектор Agilent 5793N работал в режиме полного сканирования при m/z 19 - m/z 250. В этих условиях, каждый стандарт в концентрации 100 мкг/л давал тот же самый детектируемый ответ в пределах экспериментальной ошибки.
IV. Композиции, содержащие изопрен, адсорбированный на твердой фазе
Биологически продуцируемый изопрен адсорбировали на активированном угле и получали твердую фазу, содержащую 50-99,9% углерода, 0,1%-50% изопрена, 0,01%-5% воды и небольшие количества (<0,1%) других летучих органических компонентов.
Отходящий газ, образующийся в процессе ферментации, пропускали через медный охлаждающий змеевик, поддерживаемый при 0°С, а затем этот газ пропускали через фильтр-осушитель на гранулированной двуокиси кремния для удаления водяного пара. Затем обезвоженный отходящий газ пропускали через углерод-содержащие фильтры (Koby Jr, Koby Filters, MA) до уровня, при котором проходящий поток изопрена детектировался на фильтре с вытяжным устройством при ГХ/МС. Количество изопрена, адсорбируемого на картридже, может быть определено непосредственно путем вычисления концентрации отходящего газа, общей скорости потока и процента потока, проходящего за время сбора газа. Альтернативно, адсорбированный изопрен может быть регенерирован на фильтрах посредством тепловой десорбции, вакуумной десорбции или десорбции, опосредуемой растворителем.
V. Сбор и анализ конденсированного изопрена
Отходящий газ при ферментации обезвоживали, и CO2 удаляли путем фильтрации через подходящее адсорбирующее вещество (например, аскарит). Образующийся поток отходящего газа пропускали через конденсатор с охлаждением жидким азотом для конденсации VОС в данном потоке. Сосуд для сбора содержал трет-бутилкатехол, используемый для ингибирования полученного конденсата изопрена. Конденсат анализировали с помощью СХ/МС и ЯМР для определения чистоты стандартными методами, такими как методы, описанные в настоящей заявке.
VI. Продуцирование прениловых спиртов путем ферментации
Анализ отходящего газа из штамма клеток BL21 (DE3) E.coli, экспрессирующих изопренсинтазу пуэрарии, выявил присутствие изопрена и 3-метил-3-бутен-1-ола (изопренола). Уровни этих двух соединений в отходящем газе, продуцируемом в процессе ферментации, указаны на фиг. 89 и были определены с помощью ГХ/МС в зоне «хэдспейс». В этом эксперименте, уровни продуцируемого изопренола (3-метил-3-бутен-1-ола, 3-MBA) составляли почти 10 мкг/лoтх.газа. В дополнительных экспериментах, уровни продуцируемых соединений в отходящем газе при ферментации составляли приблизительно 20 мкг/лoтх.газа.
ПРИМЕР 11: Нарушение взаимосвязи между клеточным ростом и продуцированием изопрена в клетках E.coli, экспрессирующих гены пути мевалоновой кислоты и подвергаемых ферментации в периодической культуре с подпиткой
В примере 11 проиллюстрировано нарушение взаимосвязи между клеточным ростом и продуцированием мевалоновой кислоты и изопрена.
I. Условия ферментации
Состав среды (на литр сбраживаемой среды):
Среду получали с использованием нижеследующих компонентов на литр сбраживаемой среды: 7,5 г K2HPO4, 2 г МgSO4 · 7 Н2О, 2 г моногидрата лимонной кислоты, 0,3 г цитрата железа(3)-аммония, 0,5 г дрожжевого экстракта, 1 мл 1000 × модифицированного раствора следовых количеств металла. Все компоненты добавляли одновременно и растворяли в diH2O. Этот раствор автоклавировали. рН доводили до 7,0 добавлением гидроксида аммония (30%) и объем доводили до нужной величины. После стерилизации и коррекции рН добавляли 10 г глюкозы, 0,1 г тиамина · HCl и антибиотики.
1000 × модифицированный раствор следовых количеств металла:
1000 × модифицированный раствор следовых количеств металла получали с использованием нижеследующих компонентов: 40 г лимонной кислоты · Н2О; 30 г МnSO4 · Н2О; 10 г NaCl, 1 г FeSO4 · 7 Н2О; 1 г CoCl2 · 6 Н2О, 1 г ZnSO · 7 Н2О, 100 мг CuSO4 · 5 Н2О, 100 мг H3ВО3, 100 мг NaMoO4 · 2 Н2О. Каждый компонент сразу растворяли в diН2О, рН доводили до 3,0 добавлением HCl/NaOH, а затем объем доводили до нужной величины и стерилизовали на фильтре 0,22 микрон.
Ферментацию осуществляли с использованием клеток E.coli, содержащих плазмиды pTrcHis2AUpperPathway (также обозначаемой pTrcUpperMVA, фиг. 91 и 92A-92C) (50 мкг/мл карбенициллина) или плазмиды pCL PtrcUpperMVA (также обозначаемой pCL PtrcUpperPathway (фиг. 26)) (50 мкг/мл спектиномицина). В экспериментах, в которых продуцировался изопрен, клетки E.coli также содержали плазмиду pTrcKKDyIkIS (50 мкг/мл канамицина). Эти эксперименты проводили для мониторинга образования мевалоновой кислоты или изопрена из глюкозы при нужном рН ферментации, рН=7,0, и при температуре 30°С. Инокулят штамма E.coli, взятый из замороженного сосуда, высевали штрихом на агаровый планшет с бульоном LB (в присутствии антибиотиков) и инкубировали при 37°С. Одну колонию инокулировали в среду, содержащую триптон - дрожжевой экстракт. После культивирования инокулята до оптической плотности 1,0, измеренной на 550 нм, этот инокулят использовали для инокуляции биореактора.
Глюкозу подавали с экспоненциальной скоростью до тех пор, пока клетки не достигали стационарной фазы. После этого, скорость подачи глюкозы снижали так, чтобы это удовлетворяло метаболическим требованиям. Индуцирование достигалось путем добавления IPTG. Концентрацию мевалоновой кислоты в сбраживаемом бульоне определяли путем нанесения образцов, обработанных перхлорной кислотой (Sigma-Aldrich # 244252) (0,3 M образцов, инкубированных при 4°C в течение 5 минут) на ВЭЖХ-колонку с органическими кислотами (BioRad # 125-0140). Концентрацию определяли путем сравнения высоты пика мевалоновой кислоты в бульоне в соответствии с калибровочной кривой, полученной для мевалонолактона (Sigma-Aldrich # M4667), обработанного перхлорной кислотой с образованием D,L-мевалоната. Уровень изопрена в отходящем газе биореактора определяли, как описано в настоящей заявке. Титр изопрена определяли, как количество продуцируемого изопрена на литр сбраживаемого бульона.
II. Продуцирование мевалоновой кислоты из клеток BL21 (DE3) E.coli, экспрессирующих плазмиду pTrcUpperMVA, в 150-литровом ферментере
Клетки BL21 (DE3), которые культивировали на планшете, как описано выше в примере 11, части I, инокулировали в колбу, содержащую 45 мл среды, включающей триптон-дрожжевой экстракт, и инкубировали при 30°С со встряхиванием при 170 об/мин в течение 5 часов. Это раствор переносили в 5-литровый биореактор со средой, содержащей триптон-дрожжевой экстракт, и клетки культивировали при 30°С и при 27,5 об/мин до тех пор, пока культура не достигала OD550=1,0. 5 литров инокулята высевали в 150-литровый биореактор, содержащий 45 кг среды. Когда OD550 достигала 10, концентрацию IPTG доводили до 1,1 мМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 60А. В процессе ферментации, титр мевалоновой кислоты увеличивался до конечной величины 61,3 г/л (фиг. 60В). Профиль удельной продуктивности в процессе ферментации показан на фиг. 60C, а на фиг. 60A проиллюстрировано нарушение взаимосвязи между ростом клеток и продуцированием мевалоновой кислоты. Общее количество мевалоновой кислоты, продуцируемой из 14,1 кг утилизованной глюкозы в процессе 52,5-часовой ферментации, составляло 4,0 кг. Молярный выход утилизованного углерода, который превращался в мевалоновую кислоту в процессе ферментации, составлял 34,2%.
III. Продуцирование мевалоновой кислоты из клеток BL21 (DE3) E.coli, экспрессирующих плазмиду pTrcUpperMVA, в 15-литровом ферментере
Клетки BL21 (DE3), которые культивировали на планшете, как описано выше в примере 11, части I, инокулировали в колбу, содержащую 500 мл среды, включающей триптон-дрожжевой экстракт, и культивировали при 30°С и при 160 об/мин до OD550=1,0. Этот материал высевали в 15-литровый биореактор, содержащий 4,5 кг среды. Когда OD550 достигала 10, концентрацию IPTG доводили до 1,0 мМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 61А. В процессе ферментации, титр мевалоновой кислоты увеличивался до конечной величины 53,9 г/л (фиг. 61В). Профиль удельной продуктивности в процессе ферментации показан на фиг. 61C, а на фиг. 61A проиллюстрировано нарушение взаимосвязи между ростом клеток и продуцированием мевалоновой кислоты. Общее количество мевалоновой кислоты, продуцируемой из 2,1 кг утилизованной глюкозы в процессе 46,6-часовой ферментации, составляло 491 г. Молярный выход утилизованного углерода, который превращался в мевалоновую кислоту в процессе ферментации, составлял 28,8%.
IV. Продуцирование мевалоновой кислоты из клеток FM5 E.coli, экспрессирующих плазмиду pTrcUpperMVA, в 15-литровом ферментере
Клетки FM5, которые культивировали на планшете, как описано выше в примере 11, части I, инокулировали в колбу, содержащую 500 мл среды, включающей триптон-дрожжевой экстракт, и культивировали при 30°С и при 160 об/мин до OD550=1,0. Этот материал высевали в 15-литровый биореактор, содержащий 4,5 кг среды. Когда OD550 достигала 30, концентрацию IPTG доводили до 1,0 мМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 62А. В процессе ферментации, титр мевалоновой кислоты увеличивался до конечной величины 23,7 г/л (фиг. 62В). Профиль удельной продуктивности в процессе ферментации показан на фиг. 62C, а на фиг. 62A проиллюстрировано нарушение взаимосвязи между ростом клеток и продуцированием мевалоновой кислоты. Общее количество мевалоновой кислоты, продуцируемой из 1,1 кг утилизованной глюкозы в процессе 51,2-часовой ферментации, составляло 140 г. Молярный выход утилизованного углерода, который превращался в мевалоновую кислоту в процессе ферментации, составлял 15,2%.
V. Продуцирование изопрена из клеток BL21 (DE3) E.coli, экспрессирующих плазмиды pCL PtrcUpperMVA и pTrc KKDyIkIS, в 15-литровом ферментере
Клетки BL21 (DE3), которые экспрессируют плазмиды pCL PtrcUpperMVA и pTrc KKDyIkIS, и которые культивировали на планшете, как описано выше в примере 11, части I, инокулировали в колбу, содержащую 500 мл среды, включающей триптон-дрожжевой экстракт, и выращивали при 30°С и при 160 об/мин до OD550=1,0. Этот материал высевали в 15-литровый биореактор, содержащий 4,5 кг среды. Когда OD550 достигала 10, концентрацию IPTG доводили до 25 мкМ. Когда OD550 достигала 190, концентрацию IPTG доводили до 50 мкМ. В течение 38-чаовой ферментации, концентрация IPTG увеличивалась до 100 мкМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 63А. В процессе ферментации, титр изопрена увеличивался до конечной величины 2,2 г/л бульона (фиг. 63В). Профиль удельной продуктивности в процессе ферментации показан на фиг. 63C, а на фиг. 63A проиллюстрировано нарушение взаимосвязи между ростом клеток и продуцированием изопрена. Общее количество изопрена, продуцируемого из 2,3 кг утилизованной глюкозы в процессе 54,4-часовой ферментации, составляло 15,9 г. Молярный выход утилизованного углерода, который превращался в изопрен в процессе ферментации, составлял 1,53%.
VI. Продуцирование изопрена из клеток-«переключателей» BL21 (DE3) E.coli, экспрессирующих плазмиды pCL PtrcUpperMVA и pTrc KKDyIkIS, в 15-литровом ферментере
Клетки-«переключатели» BL21 (DE3), которые экспрессируют плазмиды pCL PtrcUpperMVA и pTrc KKDyIkIS, и которые культивировали на планшете, как описано выше в примере 11, части I, инокулировали в колбу, содержащую 500 мл среды, включающей триптон-дрожжевой экстракт, и выращивали при 30°С и при 160 об/мин до OD550=1,0. Этот материал высевали в 15-литровый биореактор, содержащий 4,5 кг среды. Когда OD550 достигала 10, концентрацию IPTG доводили до 26 мкМ. Когда OD550 достигала 175, концентрацию IPTG доводили до 50 мкМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 64А. В процессе ферментации, титр изопрена увеличивался до конечной величины 1,3 г/л бульона (фиг. 64В). Профиль удельной продуктивности в процессе ферментации показан на фиг. 64C, а на фиг. 64A проиллюстрировано нарушение взаимосвязи между ростом клеток и продуцированием изопрена. Общее количество изопрена, продуцируемого из 1,6 кг утилизованной глюкозы в процессе 48,6-часовой ферментации, составляло 9,9 г. Молярный выход утилизованного углерода, который превращался в изопрен в процессе ферментации, составлял 1,34%.
VII. Продуцирование изопрена из клеток MG1655 E.coli, экспрессирующих плазмиды pCL PtrcUpperMVA и pTrc KKDyIkIS, в 15-литровом ферментере
Клетки MG1655, которые экспрессируют плазмиды pCL PtrcUpperMVA и pTrc KKDyIkIS, и которые культивировали на планшете, как описано выше в примере 11, части I, инокулировали в колбу, содержащую 500 мл среды, включающей триптон-дрожжевой экстракт, и выращивали при 30°С и при 160 об/мин до OD550=1,0. Этот материал высевали в 15-литровый биореактор, содержащий 4,5 кг среды. Когда OD550 достигала 45, концентрацию IPTG доводили до 24 мкМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 65А. В процессе ферментации, титр изопрена увеличивался до конечной величины 393 мг/л бульона (фиг. 65В). Профиль удельной продуктивности в процессе ферментации показан на фиг. 65C, а на фиг. 65A проиллюстрировано нарушение взаимосвязи между ростом клеток и продуцированием изопрена. Общее количество изопрена, продуцируемого из 520 г утилизованной глюкозы в процессе 67,4-часовой ферментации, составляло 2,2 г. Молярный выход утилизованного углерода, который превращался в изопрен в процессе ферментации, составлял 0,92%.
VIII. Продуцирование изопрена из клеток MG1655ack-pta E.coli, экспрессирующих плазмиды pCL PtrcUpperMVA и pTrc KKDyIkIS, в 15-литровом ферментере
Клетки MG1655ack-pta, которые экспрессируют плазмиды pCL PtrcUpperMVA и pTrc KKDyIkIS, и которые культивировали на планшете, как описано выше в примере 11, части I, инокулировали в колбу, содержащую 500 мл среды, включающей триптон-дрожжевой экстракт, и выращивали при 30°С и при 160 об/мин до OD550=1,0. Этот материал высевали в 15-литровый биореактор, содержащий 4,5 кг среды. Когда OD550 достигала 10, концентрацию IPTG доводили до 30 мкМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 66А. В процессе ферментации, титр изопрена увеличивался до конечной величины 368 мг/л бульона (фиг. 66В). Профиль удельной продуктивности в процессе ферментации показан на фиг. 66C, а на фиг. 66A проиллюстрировано нарушение взаимосвязи между ростом клеток и продуцированием изопрена. Общее количество изопрена, продуцируемого из 531 г утилизованной глюкозы в процессе 56,7-часовой ферментации, составляло 1,8 г. Молярный выход утилизованного углерода, который превращался в изопрен в процессе ферментации, составлял 0,73%.
IX. Продуцирование изопрена из клеток FM5 E.coli, экспрессирующих плазмиды pCL PtrcUpperMVA и pTrc KKDyIkIS, в 15-литровом ферментере
Клетки FM5, которые экспрессируют плазмиды pCL PtrcUpperMVA и pTrc KKDyIkIS, и которые культивировали на планшете, как описано выше в примере 11, части I, инокулировали в колбу, содержащую 500 мл среды, включающей триптон-дрожжевой экстракт, и выращивали при 30°С и при 160 об/мин до OD550=1,0. Этот материал высевали в 15-литровый биореактор, содержащий 4,5 кг среды. Когда OD550 достигала 15, концентрацию IPTG доводили до 27 мкМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 67А. В процессе ферментации, титр изопрена увеличивался до конечной величины 235 мг/л бульона (фиг. 67В). Профиль удельной продуктивности в процессе ферментации показан на фиг. 67C, а на фиг. 67A проиллюстрировано нарушение взаимосвязи между ростом клеток и продуцированием изопрена. Общее количество изопрена, продуцируемого из 948 г утилизованной глюкозы в процессе 52,3-часовой ферментации, составляло 1,4 г. Молярный выход утилизованного углерода, который превращался в изопрен в процессе ферментации, составлял 0,32%.
ПРИМЕР 12: Продуцирование изопрена в экспоненцальной фазе роста клеток E.coli, экспрессирующих гены пути мевалоновой кислоты и ферментированных в периодической культуре с подпиткой
В примере 12 проиллюстрировано продуцирование изопрена в экспоненцальной фазе роста клеток.
Состав среды (на литр сбраживаемой среды):
Среду получали с использованием нижеследующих компонентов на литр сбраживаемой среды: 7,5 г K2HPO4, 2 г МgSO4 · 7 Н2О, 2 г моногидрата лимонной кислоты, 0,3 г цитрата железа(3)-аммония, 0,5 г дрожжевого экстракта, 1 мл 1000 × модифицированного раствора следовых количеств металла. Все компоненты добавляли одновременно и растворяли в diH2O. Этот раствор автоклавировали. рН доводили до 7,0 добавлением гидроксида аммония (30%) и объем доводили до нужной величины. После стерилизации и коррекции рН добавляли 10 г глюкозы, 0,1 г тиамина · HCl и антибиотики.
1000 × модифицированный раствор следовых количеств металла:
1000 × модифицированный раствор следовых количеств металла получали с использованием нижеследующих компонентов: 40 г лимонной кислоты · Н2О; 30 г МnSO4 · Н2О; 10 г NaCl, 1 г FeSO4 · 7 Н2О; 1 г CoCl2 · 6 Н2О, 1 г ZnSO · 7 Н2О, 100 мг CuSO4 · 5 Н2О, 100 мг H3ВО3, 100 мг NaMoO4 · 2 Н2О. Каждый компонент сразу растворяли в diН2О, рН доводили до 3,0 добавлением HCl/NaOH, а затем объем доводили до нужной величины и стерилизовали на фильтре 0,22 микрон.
Ферментацию осуществляли в 15-литровом биореакторе с клетками E.coli АТСС11303, содержащими плазмиды pCL PtrcUpperMVA и pTrcKKDyIkIS. Этот эксперимент проводили для мониторинга образования изопрена из глюкозы при нужном рН ферментации, рН=7,0, и при температуре 30°С. Инокулят штамма E.coli, взятый из замороженного сосуда, высевали штрихом на агаровый планшет с бульоном LB (в присутствии антибиотиков) и инкубировали при 37°С. Одну колонию инокулировали в среду, содержащую триптон - дрожжевой экстракт. После культивирования инокулята до OD=1,0, измеренной на 550 нм, 500 мл этого инокулята использовали для инокуляции 5-литрового биореактора.
Глюкозу подавали с экспоненциальной скоростью до тех пор, пока клетки не достигали стационарной фазы. После этого, скорость подачи глюкозы снижали так, чтобы это удовлетворяло метаболическим требованиям. Общее количество глюкозы, подаваемое в биореактор в процессе 50-часовой ферментации, составляло 2,0 кг. Индуцирование достигалось путем добавления IPTG. Когда оптическая плотность на 550 нм (OD550) достигала значения 10, концентрацию IPTG доводили до 25 мкМ. Когда OD550 достигала 190, концентрацию IPTG доводили до 50 мкМ. Профиль OD550 в биореакторе на протяжении всего периода времени показан на фиг. 99. Уровень изопрена в отходящем газе биореактора определяли, как описано в настоящей заявке. В процессе ферментации, титр изопрена увеличивался до конечной величины 1,4 г/л (фиг. 100). Общее количество изопрена, продуцируемое в течение 50-часовой ферментации, составляло 10,0 г. Профиль удельной продуцируемости изопрена в биореакторе в течение определенного периода времени представлен на фиг. 101. Молярный выход утилизованного углерода, который превращался в изопрен в процессе ферментации, составлял 1,1%. Выход изопрена в процентах по массе глюкозы составлял 0,5%.
ПРИМЕР 13: Моделирование и испытание изопрена на воспламеняемость
I. Краткое описание моделирования воспламеняемости и испытания на воспламеняемость изопрена
Моделирование и эксперименты по воспламеняемости проводили для различных смесей углеводорода/кислорода/азота/воды/диоксида углерода. Такое моделирование и экспериментальное испытание проводили в целях построения и кривых воспламеняемости изопрена и кислорода/азота при конкретных концентрациях пара и монооксида углерода и при фиксированном давлении и температуре. Матричная схема условий моделирования представлена в таблице 4, а матричная схема проведения экспериментов представлена в таблице 5.
(°С)
(°С)
(фунт/кв. дюйм)
II. Описание модели вычисления температуры при адиабатическом воспламенении (CAFT)
В целях построения огибающей кривой воспламенения изопрена вычисляли температуры при адиабатическом воспламенении (CAFT), а также проводили оценку выбранного температурного предела воспламеняемости для анализа распространения пламени. В этом исследовании, для вычисления температур воспламеняемости использовали компьютерную программу NASA Glenn Research Center CEA (программу оценки химического равновесия с приложениями).
Построение огибающей кривой воспламеняемости с использованием модели температуры при адиабатическом воспламенении для механизма гомогенного горения (где топливо и окислитель присутствуют в газообразном состоянии) проводили в пять стадий: выбора нужных реагентов, выбора условий испытания, выбора температурного предела воспламеняемости, модификации реагентов и построения огибающей кривой воспламеняемости на основе этих вычислений.
В этой первой стадии выбора нужных реагентов следует принять решение, какие именно реагенты должны присутствовать в системе и каким должно быть количество каждого из них. Во многих случаях, в компьютерных программах, используемых для таких вычислений, имеется список реагентов и продуктов различных видов. Если в программе отсутствуют какие-либо данные об исследуемых видах реагентов и продуктов, то они могут быть взяты из других источников, таких как таблицы JANAF, или из интернета. В этой современной модели, в компьютерной базе данных имеются данные для воды, азота, кислорода и диоксида углерода. В этой компьютерной базе данных отсутствует изопрен как вид, а поэтому термодинамические свойства вводят вручную.
В следующей стадии необходимо решить, происходит ли процесс горения при начальном давлении и начальной температуре. В этой модели, давление составляет 1 атмосферу (абсолютное давление), а температура составляет 40°С, то есть точка кипения изопрена.
Предел температуры воспламенения для горения может быть выбран либо теоретически, либо экспериментально. Каждый метод имеет свои собственные ограничения.
Исходя из предыдущих исследований, предел температуры воспламенения углеводородов составляет от 1000 K до 1500 K. Для этой модели была выбрана величина 1500 K. При этой температуре, реакция превращения монооксида углерода в диоксид углерода (в высокой степени экзотермическая реакция, которая потребляет значительную часть энергии пламени) поддерживается самостоятельно.
После определения предела температуры воспламенения, проводили вычисление модели исходя из данной смеси реагентов (концентраций молекул) и определяли температуру при адиабатическом воспламенении. Считается, что распространение пламени происходит только в том случае, если температура выше предела температуры воспламенения. Затем состав смеси реагентов модифицировали в целях для получения набора данных для реакционноспособных и нереакционноспособных смесей.
Модель этого типа показала хорошее соответствие с экспериментально определенными пределами воспламеняемости. Области, находящиеся за пределами дифференциальной огибающей кривой, являются невоспламеняемыми, а области, находящиеся в пределах такой кривой, являются воспламеняемыми. Эта огибающая кривая имеет форму «носа». «Нос» огибающей кривой соответствует предельной концентрации кислорода (LOC) для газообразного топлива.
III. Результаты модели вычисления температур при адиабатическом воспламенении (CAFT)
На графиках, представленных на фиг. 68-74, приводятся результаты вычисления модели CAFT для серий A-G, соответственно. На этих фигурах представлены графики зависимости вычисленной температуры при адиабатическом воспламенении (с использованием программы NASA CEA) от концентрации топлива (по массе) для некоторых отношений кислород/азот (по массе). Участки кривой, которые находятся выше выбранного предела температуры воспламенения 1500 K, соответствуют уровням топлива, достаточным для распространения пламени. Эти результаты трудно поддаются интерпретации в форме, представленной на фиг. 68-74. Кроме того, такая используемая в настоящее время форма не позволяет проводить сравнение экспериментальных данных, которые обычно представлены в процентах по объему.
При использовании серии A, взятой в качестве примера, данные на фиг. 68 могут быть представлены в форме традиционной огибающей кривой воспламеняемости. С использованием фиг. 68 и по линии, проходящей от значения на ординате, соответствующего температуре 1500 К, можно определить концентрацию топлива для этого предела температуры воспламенения путем проведения линии от точки касания к абсциссе для каждой кривой (отношение кислорода к азоту), которая пересекает эту линию. Полученные величины могут быть затем представлены в табличной форме как масс.% топлива для данного масс.% окислителя (фиг. 75A). После определения состава топлива (100 масс.% изопрена) и состава окислителя (относительное содержание воды, кислорода и азота) можно вычислить молярные количества.
Исходя из этих молярных количеств можно вычислить концентрации в процентах по объему. Затем, исходя из концентраций в процентах по объему, может быть построена огибающая кривая воспламеняемости (фиг. 75B). Площадь, ограниченная огибающей кривой, входит в зону взрываемости, а область за пределами этой кривой входит в зону отсутствия взрываемости. «Нос» огибающей кривой соответствует предельной концентрации кислорода. На фиг. 76A и 76B представлены вычисленные концентрации по объему для огибающей кривой воспламеняемости для серии В, построенной по данным, приведенным на фиг. 69. Аналогичный подход может быть применен для данных, представленных на фиг. 70-74.
IV. Оборудование для экспериментального испытания на воспламеняемость и процедура проведения этого испытания
Испытание на воспламеняемость проводили в 4-литровом сосуде высокого давления. Этот сосуд имеет цилиндрическую форму с внутренним диаметром 6" и внешней высотой 8,625". Температуру сосуда (и находящихся внутри газов) поддерживали с помощью внешних нагревателей, снабженных ПИД-регулятором (пропорционально-интегрально-дифференциальным регулятором). Для предупреждения потери тепла, сосуд под давлением был обернут керамической шерстью и отражающим изоляционным материалом. Для измерения температуры заполненного газом пространства, а также температуры самого сосуда использовали термопары типа К. Сосуд для проведения такого испытания показан на фиг. 77.
Перед проведением данного испытания, из сосуда откачивали воздух, и этот сосуд продували азотом для гарантии удаления всех газов, образовавшихся при проведении предыдущих испытаний. Затем в сосуд подавали вакуум. После этого давление обычно составляло примерно 0,06 бар. Поскольку продувку проводили азотом, то, очевидно, что газом, создающим такое начальное давление, является азот. Затем, с использованием парциального давления, в этот сосуд подавали соответствующие количества воды, изопрена, азота и кислорода для обеспечения требуемых условий испытания. В сосуд был установлен магнитно-индукционный вентилятор для смешивания газообразного содержимого. Газы смешивали этим вентилятором в течение примерно 2 минут, а приблизительно за 1 минуту до вынужденного воспламенения, вентилятор отключали.
Воспламенитель состоял из нихромового змеевика с сопротивлением 1,5 Ом и источника переменного напряжения (VAC), подсоединенного к таймеру. С использованием осциллоскопа было определено, что VAC=34,4 подается на воспламенитель за 3,2 секунды. Максимальный ток в 3,8 Ампер достигается примерно в середине цикла воспламенения. Таким образом, максимальная мощность составляет 131 Ватт, а общая энергия во время цикла воспламенения составляет приблизительно 210 Дж.
Данные по мгновенному сгоранию были получены с использованием магнитоэлектрического измерительного преобразователя давления Validyne DP215, подсоединенного к системе сбора данных. При этом считается, что газовая смесь мгновенно сгорает, если давление превышает или равно 5%.
V. Результаты испытаний на воспламенение
Первую серию экспериментов (серию 1) проводили при температуре 40°С и давлении 0 фунт/кв. дюйм в отсутствии пара. После проведения испытаний при различных концентрациях изопрена и кислорода строили кривую воспламеняемости, представленную на фиг.78А. Исходными данными, представленными на этой кривой, являются лишь данные, ограничивающие данную кривую. Подробный список всех исходных данных, взятых для этой серии, приводится на фиг. 80A и 80B.
На фиг. 78B систематизированы исходные данные по взрываемости, представленные на фиг.78А. На фиг.78С проиллюстрировано сравнение экспериментальных данных с данными, предсказанными по огибающей кривой воспламеняемости для модели CAFT. Эта модель в полной мере соответствует экспериментальным данным. Некоторые расхождения в данных можно объяснить не-адиабатическими условиями камеры для испытаний и ограничениями самой модели. Эта модель предусматривает неограниченный временной горизонт для реакции окисления и не учитывает каких-либо кинетических ограничений реакции.
Кроме того, эта модель ограничена числом равновесных химических молекул, которые имеются в базе данных, а поэтому такая модель не может точно предсказать пиролитические процессы. Кроме того, в огибающей кривой, построенной по этой модели, используется одно значение предела температуры воспламенения (1500K). Предел температуры воспламенения может составлять от 1000K до 1500K в зависимости от реакционной способности химических молекул. Одной из причин, по которой такая модель не может точно предсказать верхний предел воспламеняемости для этой системы, является сложная природа пиролитических химических молекул, образующихся в том случае, когда концентрация топлива превышает стехиометрическую концентрацию топлива/окислителя.
Вторую серию экспериментов (серию 2) проводили при температуре 40°С и давлении 0 фунт/кв. дюйм при фиксированной концентрации пара 4%. После проведения испытаний при различных концентрациях изопрена и кислорода строили кривую воспламеняемости, представленную на фиг.79А. Исходными данными, представленными на этой кривой, являются лишь данные, ограничивающие данную кривую. Подробный список всех исходных данных, взятых для этой серии, приводится на фиг. 81. Из-за сходства с данными серии 1 были протестированы лишь ключевые величины нижнего предела воспламеняемости, величины предельной концентрации кислорода и величины верхнего предела воспламеняемости. Добавление 4% пара в испытуемую смесь не приводило к значительному изменению ключевых пределов огибающей кривой воспламеняемости. При этом, следует отметить, что более высокие концентрации пара/воды и/или других инертных веществ могут оказывать влияние на огибающую кривую воспламеняемости.
На фиг. 79B систематизированы исходные данные по взрываемости, представленные на фиг. 79А. На фиг. 79С проиллюстрировано сравнение экспериментальных данных с данными, предсказанными по огибающей кривой воспламеняемости для модели CAFT. Эта модель в полной мере соответствует экспериментальным данным. Некоторые расхождения в данных можно объяснить теми же самыми факторами, приведенными выше в описании экспериментов серии 1.
V. Вычисление пределов воспламеняемости изопрена в воздухе под давлением в 3 атмосферы
Методы, описанные в примере 13, частях I-IV, были также применены для вычисления пределов воспламеняемости изопрена при абсолютном давлении системы в 3 атмосферы и при температуре 40°С. Полученные результаты сравнивали с результатами примера 13, частей I-IV, полученными при абсолютном давлении системы в 1 атмосферу и при температуре 40°С. Были проведены испытания при высоком давлении, поскольку огибающая кривая воспламеняемости расширяется или увеличивается по мере повышения начального давления системы. На верхний предел воспламеняемости влияет множество факторов, а также ограничение содержания кислорода. На нижний предел воспламеняемости влияет меньшее число факторов (см., например, публикацию «Bulletin 627-Flammability Characteristics of Combustible Gases and Vapors», Michael G. Zabetakis, опубликованную ранее Горным Управлением США (1965), которая во всей своей полноте вводится в настоящее описание посредством ссылки, а в частности, в описание, относящееся к вычислениям пределов воспламеняемости).
На фигуре 82, вычисленная температура при адиабатическом воспламенении зависит от концентрации изопрена (топлива), выраженной в масс.% от общего содержания топлива/азота/кислорода, где начальное давление системы составляет 3 атмосферы. Вычисленные температуры воспламенения имеют большое сходство с определенными ранее температурами при давлении системы в 1 атмосферу (фиг. 83). В результате этого было обнаружено, что при построении огибающих кривых воспламеняемости с использованием вычисленных данных адиабатического воспламенения, эти кривые имеют большое сходство (см. фиг. 84 и 85). Поэтому, на основании этих теоретических вычислений был сделан вывод, что повышение давления системы от 1 атмосферы до 3 атмосфер не приводит к значительному увеличению/расширению огибающей кривой воспламеняемости. При желании, результаты этой модели могут быть подтверждены путем экспериментального испытания (такого, как описанное здесь экспериментальное испытание, проводимое под давлением в 1 атмосферу).
VII. Краткое заключение по исследованиям воспламеняемости
Вычисленная модель для температуры при адиабатическом воспламенении была разработана для построения огибающей кривой воспламеняемости системы изопрена/кислорода/азота/воды/диоксида углерода при температуре 40°С и давлении 0 фунт/кв. дюйм. Разработанная модель CAFT точно соответствует экспериментальным данным, полученным путем проведения испытаний, описанных в этой работе. Экспериментальные данные серии 1 и 2 подтвердили результаты для модели серии A и B.
Если это конкретно не определено в описании настоящей заявки, то все используемые здесь технические и научные термины имеют значения, в основном, понятные любому среднему специалисту в области, к которой относится настоящее изобретение. Так, например, в публикациях Singleton at al., Dictionary of Microbiology and Molecular Biology, 2d Ed., John Wiley and Sons, NY (1994); и Hale and Marham, The Harper Collins Dictionary of Biology, Harper Perennial, NY (1991), а также в общих словарях имеется много терминов, обычно употребляемых специалистами и используемых в настоящем изобретении. Следует отметить, что настоящее изобретение не ограничивается конкретными описанными здесь методами, протоколами и реагентами, которые могут варьироваться. Для специалиста в данной области очевидно, что для практического осуществления настоящего изобретения или для проведения испытаний, описанных в настоящей заявке, могут быть применены любые методы и материалы, аналогичные или эквивалентные описанным здесь методам и материалам.
Кроме того, представленные в настоящей заявке заголовки разделов не ограничивают различные аспекты или варианты настоящего изобретения, которые упоминаются в настоящей заявке посредством ссылки на описание в целом.
В настоящем описании, при употреблении артиклей «а», «an» и «the» подразумевается, что существительные, с которыми употребляются эти артикли, могут присутствовать как в единственном числе, так и во множественном числе, если это не оговорено особо.
Используемый здесь термин «приблизительно», относящийся к величине или параметру, включает (и описывает) изменения, которые были внесены в данные величины или параметр per se. Так, например, термин «примерно X» включает «Х». Численные интервалы включают все числа, определяющие данный численный интервал.
Следует отметить, что настоящее изобретение, в котором описаны конкретные аспекты и варианты, «включает», «состоит» и/или «по существу, состоит из» этих аспектов и вариантов.
ПРИЛОЖЕНИЕ 1
Репрезентативные нуклеиновые кислоты и полипептиды 1-дезокси-D-ксилулозо-5-фосфатсинтазы
ATH: http://www.genome.ad.jp/dbget-bin/www_bget?ath+AT3G21500(DXPS1) http://www.genome.ad.jp/dbget-bin/www_bget?ath+AT4G15560(CLA1) http://www.genome.ad.jp/dbget-bin/www_bget?ath+AT5G11380(DXPS3)
OSA: http://www.genome.ad.jp/dbget-bin/www_bget?osa+4338768 http://www.genome.ad.jp/dbget-bin/www_bget?osa+4340090 http://www.genome.ad.jp/dbget-bin/www_bget?osa+4342614
CME: http://www.genome.ad.jp/dbget-bin/www_bget?cme+CMF089C
PFA: http://www.genome.ad.jp/dbget-bin/www_bget?pfa+MAL13P1.186
TAN: http://www.genome.ad.jp/dbget-bin/www_bget?tan+TA20470
TPV: http://www.genome.ad.jp/dbget-bin/www_bget?tpv+TP01_0516
ECO: http://www.genome.ad.jp/dbget-bin/www_bget?eco+b0420(dxs)
ECJ: http://www.genome.ad.jp/dbget-bin/www_bget?ecj+JW0410(dxs)
ECE: http://www.genome.ad.jp/dbget-bin/www_bget?ece+Z0523(dxs)
ECS: http://www.genome.ad.jp/dbget-bin/www_bget?ecs+ECs0474
ECC: http://www.genome.ad.jp/dbget-bin/www_bget?ecc+c0531(dxs)
ECI: http://www.genome.ad.jp/dbget-bin/www_bget?eci+UTI89_C0443(dxs)
ECP: http://www.genome.ad.jp/dbget-bin/www_bget?ecp+ECP_0479
ECV: http://www.genome.ad.jp/dbget-bin/www_bget?ecv+APECO1_1590(dxs)
ECW: http://www.genome.ad.jp/dbget-bin/www_bget?ecw+EcE24377A_0451(dxs)
ECX: http://www.genome.ad.jp/dbget-bin/www_bget?ecx+EcHS_A0491
STY: http://www.genome.ad.jp/dbget-bin/www_bget?sty+STY0461(dxs)
STT: http://www.genome.ad.jp/dbget-bin/www_bget?stt+t2441(dxs)
SPT: http://www.genome.ad.jp/dbget-bin/www_bget?spt+SPA2301(dxs)
SEC: http://www.genome.ad.jp/dbget-bin/www_bget?sec+SC0463(dxs)
STM: http://www.genome.ad.jp/dbget-bin/www_bget?stm+STM0422(dxs)
YPE: http://www.genome.ad.jp/dbget-bin/www_bget?ype+YPO3177(dxs)
YPK: http://www.genome.ad.jp/dbget-bin/www_bget?ypk+y1008(dxs)
YPM: http://www.genome.ad.jp/dbget-bin/www_bget?ypm+YP_0754(dxs)
YPA: http://www.genome.ad.jp/dbget-bin/www_bget?ypa+YPA_2671
YPN: http://www.genome.ad.jp/dbget-bin/www_bget?ypn+YPN_0911
YPP: http://www.genome.ad.jp/dbget-bin/www_bget?ypp+YPDSF_2812
YPS: http://www.genome.ad.jp/dbget-bin/www_bget?yps+YPTB0939(dxs)
YPI: http://www.genome.ad.jp/dbget-bin/www_bget?ypi+YpsIP31758_3112(dxs)
SFL: http://www.genome.ad.jp/dbget-bin/www_bget?sfl+SF0357(dxs)
SFX: http://www.genome.ad.jp/dbget-bin/www_bget?sfx+S0365(dxs)
SFV: http://www.genome.ad.jp/dbget-bin/www_bget?sfv+SFV_0385(dxs)
SSN: http://www.genome.ad.jp/dbget-bin/www_bget?ssn+SSON_0397(dxs)
SBO: http://www.genome.ad.jp/dbget-bin/www_bget?sbo+SBO_0314(dxs)
SDY: http://www.genome.ad.jp/dbget-bin/www_bget?sdy+SDY_0310(dxs)
ECA: http://www.genome.ad.jp/dbget-bin/www_bget?eca+ECA1131(dxs)
PLU: http://www.genome.ad.jp/dbget-bin/www_bget?plu+plu3887(dxs)
BUC: http://www.genome.ad.jp/dbget-bin/www_bget?buc+BU464(dxs)
BAS: http://www.genome.ad.jp/dbget-bin/www_bget?bas+BUsg448(dxs)
WBR: http://www.genome.ad.jp/dbget-bin/www_bget?wbr+WGLp144(dxs)
SGL: http://www.genome.ad.jp/dbget-bin/www_bget?sgl+SG0656
KPN: http://www.genome.ad.jp/dbget-bin/www_bget?kpn+KPN_00372(dxs)
BFL: http://www.genome.ad.jp/dbget-bin/www_bget?bfl+Bfl238(dxs)
BPN: http://www.genome.ad.jp/dbget-bin/www_bget?bpn+BPEN_244(dxs)
HIN: http://www.genome.ad.jp/dbget-bin/www_bget?hin+HI1439(dxs)
HIT: http://www.genome.ad.jp/dbget-bin/www_bget?hit+NTHI1691(dxs)
HIP: http://www.genome.ad.jp/dbget-bin/www_bget?hip+CGSHiEE_04795
HIQ: http://www.genome.ad.jp/dbget-bin/www_bget?hiq+CGSHiGG_01080
HDU: http://www.genome.ad.jp/dbget-bin/www_bget?hdu+HD0441(dxs)
HSO: http://www.genome.ad.jp/dbget-bin/www_bget?hso+HS_0905(dxs)
PMU: http://www.genome.ad.jp/dbget-bin/www_bget?pmu+PM0532(dxs)
MSU: http://www.genome.ad.jp/dbget-bin/www_bget?msu+MS1059(dxs)
APL: http://www.genome.ad.jp/dbget-bin/www_bget?apl+APL_0207(dxs)
XFA: http://www.genome.ad.jp/dbget-bin/www_bget?xfa+XF2249
XFT: http://www.genome.ad.jp/dbget-bin/www_bget?xft+PD1293(dxs)
XCC: http://www.genome.ad.jp/dbget-bin/www_bget?xcc+XCC2434(dxs)
XCB: http://www.genome.ad.jp/dbget-bin/www_bget?xcb+XC_1678
XCV: http://www.genome.ad.jp/dbget-bin/www_bget?xcv+XCV2764(dxs)
XAC: http://www.genome.ad.jp/dbget-bin/www_bget?xac+XAC2565(dxs)
XOO: http://www.genome.ad.jp/dbget-bin/www_bget?xoo+XOO2017(dxs)
XOM: http://www.genome.ad.jp/dbget-bin/www_bget?xom+XOO_1900(XOO1900)
VCH: http://www.genome.ad.jp/dbget-bin/www_bget?vch+VC0889
VVU: http://www.genome.ad.jp/dbget-bin/www_bget?vvu+VV1_0315
VVY: http://www.genome.ad.jp/dbget-bin/www_bget?vvy+VV0868
VPA: http://www.genome.ad.jp/dbget-bin/www_bget?vpa+VP0686
VFI: http://www.genome.ad.jp/dbget-bin/www_bget?vfi+VF0711
PPR: http://www.genome.ad.jp/dbget-bin/www_bget?ppr+PBPRA0805
PAE: http://www.genome.ad.jp/dbget-bin/www_bget?pae+PA4044(dxs)
PAU: http://www.genome.ad.jp/dbget-bin/www_bget?pau+PA14_11550(dxs)
PAP: http://www.genome.ad.jp/dbget-bin/www_bget?pap+PSPA7_1057(dxs)
PPU: http://www.genome.ad.jp/dbget-bin/www_bget?ppu+PP_0527(dxs)
PST: http://www.genome.ad.jp/dbget-bin/www_bget?pst+PSPTO_0698(dxs)
PSB: http://www.genome.ad.jp/dbget-bin/www_bget?psb+Psyr_0604
PSP: http://www.genome.ad.jp/dbget-bin/www_bget?psp+PSPPH_0599(dxs)
PFL: http://www.genome.ad.jp/dbget-bin/www_bget?pfl+PFL_5510(dxs)
PFO: http://www.genome.ad.jp/dbget-bin/www_bget?pfo+Pfl_5007
PEN: http://www.genome.ad.jp/dbget-bin/www_bget?pen+PSEEN0600(dxs)
PMY: http://www.genome.ad.jp/dbget-bin/www_bget?pmy+Pmen_3844
PAR: http://www.genome.ad.jp/dbget-bin/www_bget?par+Psyc_0221(dxs)
PCR: http://www.genome.ad.jp/dbget-bin/www_bget?pcr+Pcryo_0245
ACI: http://www.genome.ad.jp/dbget-bin/www_bget?aci+ACIAD3247(dxs)
SON: http://www.genome.ad.jp/dbget-bin/www_bget?son+SO_1525(dxs)
SDN: http://www.genome.ad.jp/dbget-bin/www_bget?sdn+Sden_2571
SFR: http://www.genome.ad.jp/dbget-bin/www_bget?sfr+Sfri_2790
SAZ: http://www.genome.ad.jp/dbget-bin/www_bget?saz+Sama_2436
SBL: http://www.genome.ad.jp/dbget-bin/www_bget?sbl+Sbal_1357
SLO: http://www.genome.ad.jp/dbget-bin/www_bget?slo+Shew_2771
SHE: http://www.genome.ad.jp/dbget-bin/www_bget?she+Shewmr4_2731
SHM: http://www.genome.ad.jp/dbget-bin/www_bget?shm+Shewmr7_2804
SHN: http://www.genome.ad.jp/dbget-bin/www_bget?shn+Shewana3_2901
SHW: http://www.genome.ad.jp/dbget-bin/www_bget?shw+Sputw3181_2831
ILO: http://www.genome.ad.jp/dbget-bin/www_bget?ilo+IL2138(dxs)
CPS: http://www.genome.ad.jp/dbget-bin/www_bget?cps+CPS_1088(dxs)
PHA: http://www.genome.ad.jp/dbget-bin/www_bget?pha+PSHAa2366(dxs)
PAT: http://www.genome.ad.jp/dbget-bin/www_bget?pat+Patl_1319
SDE: http://www.genome.ad.jp/dbget-bin/www_bget?sde+Sde_3381
PIN: http://www.genome.ad.jp/dbget-bin/www_bget?pin+Ping_2240
MAQ: http://www.genome.ad.jp/dbget-bin/www_bget?maq+Maqu_2438
MCA: http://www.genome.ad.jp/dbget-bin/www_bget?mca+MCA0817(dxs)
FTU: http://www.genome.ad.jp/dbget-bin/www_bget?ftu+FTT1018c(dxs)
FTF: http://www.genome.ad.jp/dbget-bin/www_bget?ftf+FTF1018c(dxs)
FTW: http://www.genome.ad.jp/dbget-bin/www_bget?ftw+FTW_0925(dxs)
FTL: http://www.genome.ad.jp/dbget-bin/www_bget?ftl+FTL_1072
FTH: http://www.genome.ad.jp/dbget-bin/www_bget?fth+FTH_1047(dxs)
FTA: http://www.genome.ad.jp/dbget-bin/www_bget?fta+FTA_1131(dxs)
FTN: http://www.genome.ad.jp/dbget-bin/www_bget?ftn+FTN_0896(dxs)
NOC: http://www.genome.ad.jp/dbget-bin/www_bget?noc+Noc_1743
AEH: http://www.genome.ad.jp/dbget-bin/www_bget?aeh+Mlg_1381
HCH: http://www.genome.ad.jp/dbget-bin/www_bget?hch+HCH_05866(dxs)
CSA: http://www.genome.ad.jp/dbget-bin/www_bget?csa+Csal_0099
ABO: http://www.genome.ad.jp/dbget-bin/www_bget?abo+ABO_2166(dxs)
AHA: http://www.genome.ad.jp/dbget-bin/www_bget?aha+AHA_3321(dxs)
BCI: http://www.genome.ad.jp/dbget-bin/www_bget?bci+BCI_0275(dxs)
RMA: http://www.genome.ad.jp/dbget-bin/www_bget?rma+Rmag_0386
VOK: http://www.genome.ad.jp/dbget-bin/www_bget?vok+COSY_0360(dxs)
NME: http://www.genome.ad.jp/dbget-bin/www_bget?nme+NMB1867
NMA: http://www.genome.ad.jp/dbget-bin/www_bget?nma+NMA0589(dxs)
NMC: http://www.genome.ad.jp/dbget-bin/www_bget?nmc+NMC0352(dxs)
NGO: http://www.genome.ad.jp/dbget-bin/www_bget?ngo+NGO0036
CVI: http://www.genome.ad.jp/dbget-bin/www_bget?cvi+CV_2692(dxs)
RSO: http://www.genome.ad.jp/dbget-bin/www_bget?rso+RSc2221(dxs)
REU: http://www.genome.ad.jp/dbget-bin/www_bget?reu+Reut_A0882
REH: http://www.genome.ad.jp/dbget-bin/www_bget?reh+H16_A2732(dxs)
RME: http://www.genome.ad.jp/dbget-bin/www_bget?rme+Rmet_2615
BMA: http://www.genome.ad.jp/dbget-bin/www_bget?bma+BMAA0330(dxs)
BMV: http://www.genome.ad.jp/dbget-bin/www_bget?bmv+BMASAVP1_1512(dxs)
BML: http://www.genome.ad.jp/dbget-bin/www_bget?bml+BMA10299_1706(dxs)
BMN: http://www.genome.ad.jp/dbget-bin/www_bget?bmn+BMA10247_A0364(dxs)
BXE: http://www.genome.ad.jp/dbget-bin/www_bget?bxe+Bxe_B2827
BUR: http://www.genome.ad.jp/dbget-bin/www_bget?bur+Bcep18194_B2211
BCN: http://www.genome.ad.jp/dbget-bin/www_bget?bcn+Bcen_4486
BCH: http://www.genome.ad.jp/dbget-bin/www_bget?bch+Bcen2424_3879
BAM: http://www.genome.ad.jp/dbget-bin/www_bget?bam+Bamb_3250
BPS: http://www.genome.ad.jp/dbget-bin/www_bget?bps+BPSS1762(dxs)
BPM: http://www.genome.ad.jp/dbget-bin/www_bget?bpm+BURPS1710b_A0842(dxs)
BPL: http://www.genome.ad.jp/dbget-bin/www_bget?bpl+BURPS1106A_A2392(dxs)
BPD: http://www.genome.ad.jp/dbget-bin/www_bget?bpd+BURPS668_A2534(dxs)
BTE: http://www.genome.ad.jp/dbget-bin/www_bget?bte+BTH_II0614(dxs)
BPE: http://www.genome.ad.jp/dbget-bin/www_bget?bpe+BP2798(dxs)
BPA: http://www.genome.ad.jp/dbget-bin/www_bget?bpa+BPP2464(dxs)
BBR: http://www.genome.ad.jp/dbget-bin/www_bget?bbr+BB1912(dxs)
RFR: http://www.genome.ad.jp/dbget-bin/www_bget?rfr+Rfer_2875
POL: http://www.genome.ad.jp/dbget-bin/www_bget?pol+Bpro_1747
PNA: http://www.genome.ad.jp/dbget-bin/www_bget?pna+Pnap_1501
AJS: http://www.genome.ad.jp/dbget-bin/www_bget?ajs+Ajs_1038
MPT: http://www.genome.ad.jp/dbget-bin/www_bget?mpt+Mpe_A2631
HAR: http://www.genome.ad.jp/dbget-bin/www_bget?har+HEAR0279(dxs)
MMS: http://www.genome.ad.jp/dbget-bin/www_bget?mms+mma_0331
NEU: http://www.genome.ad.jp/dbget-bin/www_bget?neu+NE1161(dxs)
NET: http://www.genome.ad.jp/dbget-bin/www_bget?net+Neut_1501
NMU: http://www.genome.ad.jp/dbget-bin/www_bget?nmu+Nmul_A0236
EBA: http://www.genome.ad.jp/dbget-bin/www_bget?eba+ebA4439(dxs)
AZO: http://www.genome.ad.jp/dbget-bin/www_bget?azo+azo1198(dxs)
DAR: http://www.genome.ad.jp/dbget-bin/www_bget?dar+Daro_3061
TBD: http://www.genome.ad.jp/dbget-bin/www_bget?tbd+Tbd_0879
MFA: http://www.genome.ad.jp/dbget-bin/www_bget?mfa+Mfla_2133
HPY: http://www.genome.ad.jp/dbget-bin/www_bget?hpy+HP0354(dxs)
HPJ: http://www.genome.ad.jp/dbget-bin/www_bget?hpj+jhp0328(dxs)
HPA: http://www.genome.ad.jp/dbget-bin/www_bget?hpa+HPAG1_0349
HHE: http://www.genome.ad.jp/dbget-bin/www_bget?hhe+HH0608(dxs)
HAC: http://www.genome.ad.jp/dbget-bin/www_bget?hac+Hac_0968(dxs)
WSU: http://www.genome.ad.jp/dbget-bin/www_bget?wsu+WS1996
TDN: http://www.genome.ad.jp/dbget-bin/www_bget?tdn+Tmden_0475
CJE: http://www.genome.ad.jp/dbget-bin/www_bget?cje+Cj0321(dxs)
CJR: http://www.genome.ad.jp/dbget-bin/www_bget?cjr+CJE0366(dxs)
CJJ: http://www.genome.ad.jp/dbget-bin/www_bget?cjj+CJJ81176_0343(dxs)
CJU: http://www.genome.ad.jp/dbget-bin/www_bget?cju+C8J_0298(dxs)
CJD: http://www.genome.ad.jp/dbget-bin/www_bget?cjd+JJD26997_1642(dxs)
CFF: http://www.genome.ad.jp/dbget-bin/www_bget?cff+CFF8240_0264(dxs)
CCV: http://www.genome.ad.jp/dbget-bin/www_bget?ccv+CCV52592_1671(dxs) http://www.genome.ad.jp/dbget-bin/www_bget?ccv+CCV52592_1722
CHA: http://www.genome.ad.jp/dbget-bin/www_bget?cha+CHAB381_1297(dxs)
CCO: http://www.genome.ad.jp/dbget-bin/www_bget?cco+CCC13826_1594(dxs)
ABU: http://www.genome.ad.jp/dbget-bin/www_bget?abu+Abu_2139(dxs)
NIS: http://www.genome.ad.jp/dbget-bin/www_bget?nis+NIS_0391(dxs)
SUN: http://www.genome.ad.jp/dbget-bin/www_bget?sun+SUN_2055(dxs)
GSU: http://www.genome.ad.jp/dbget-bin/www_bget?gsu+GSU0686(dxs-1) http://www.genome.ad.jp/dbget-bin/www_bget?gsu+GSU1764(dxs-2)
GME: http://www.genome.ad.jp/dbget-bin/www_bget?gme+Gmet_1934 http://www.genome.ad.jp/dbget-bin/www_bget?gme+Gmet_2822
PCA: http://www.genome.ad.jp/dbget-bin/www_bget?pca+Pcar_1667
PPD: http://www.genome.ad.jp/dbget-bin/www_bget?ppd+Ppro_1191 http://www.genome.ad.jp/dbget-bin/www_bget?ppd+Ppro_2403
DVU: http://www.genome.ad.jp/dbget-bin/www_bget?dvu+DVU1350(dxs)
DVL: http://www.genome.ad.jp/dbget-bin/www_bget?dvl+Dvul_1718
DDE: http://www.genome.ad.jp/dbget-bin/www_bget?dde+Dde_2200
LIP: http://www.genome.ad.jp/dbget-bin/www_bget?lip+LI0408(dsx)
DPS: http://www.genome.ad.jp/dbget-bin/www_bget?dps+DP2700
ADE: http://www.genome.ad.jp/dbget-bin/www_bget?ade+Adeh_1097
MXA: http://www.genome.ad.jp/dbget-bin/www_bget?mxa+MXAN_4643(dxs)
SAT: http://www.genome.ad.jp/dbget-bin/www_bget?sat+SYN_02456
SFU: http://www.genome.ad.jp/dbget-bin/www_bget?sfu+Sfum_1418
PUB: http://www.genome.ad.jp/dbget-bin/www_bget?pub+SAR11_0611(dxs)
MLO: http://www.genome.ad.jp/dbget-bin/www_bget?mlo+mlr7474
MES: http://www.genome.ad.jp/dbget-bin/www_bget?mes+Meso_0735
SME: http://www.genome.ad.jp/dbget-bin/www_bget?sme+SMc00972(dxs)
ATU: http://www.genome.ad.jp/dbget-bin/www_bget?atu+Atu0745(dxs)
ATC: http://www.genome.ad.jp/dbget-bin/www_bget?atc+AGR_C_1351
RET: http://www.genome.ad.jp/dbget-bin/www_bget?ret+RHE_CH00913(dxs)
RLE: http://www.genome.ad.jp/dbget-bin/www_bget?rle+RL0973(dxs)
BME: http://www.genome.ad.jp/dbget-bin/www_bget?bme+BMEI1498
BMF: http://www.genome.ad.jp/dbget-bin/www_bget?bmf+BAB1_0462(dxs)
BMS: http://www.genome.ad.jp/dbget-bin/www_bget?bms+BR0436(dxs)
BMB: http://www.genome.ad.jp/dbget-bin/www_bget?bmb+BruAb1_0458(dxs)
BOV: http://www.genome.ad.jp/dbget-bin/www_bget?bov+BOV_0443(dxs)
BJA: http://www.genome.ad.jp/dbget-bin/www_bget?bja+bll2651(dxs)
BRA: http://www.genome.ad.jp/dbget-bin/www_bget?bra+BRADO2161(dxs)
BBT: http://www.genome.ad.jp/dbget-bin/www_bget?bbt+BBta_2479(dxs)
RPA: http://www.genome.ad.jp/dbget-bin/www_bget?rpa+RPA0952(dxs)
RPB: http://www.genome.ad.jp/dbget-bin/www_bget?rpb+RPB_4460
RPC: http://www.genome.ad.jp/dbget-bin/www_bget?rpc+RPC_1149
RPD: http://www.genome.ad.jp/dbget-bin/www_bget?rpd+RPD_4305
RPE: http://www.genome.ad.jp/dbget-bin/www_bget?rpe+RPE_1067
NWI: http://www.genome.ad.jp/dbget-bin/www_bget?nwi+Nwi_0633
NHA: http://www.genome.ad.jp/dbget-bin/www_bget?nha+Nham_0778
BHE: http://www.genome.ad.jp/dbget-bin/www_bget?bhe+BH04350(dxs)
BQU: http://www.genome.ad.jp/dbget-bin/www_bget?bqu+BQ03540(dxs)
BBK: http://www.genome.ad.jp/dbget-bin/www_bget?bbk+BARBAKC583_0400(dxs)
CCR: http://www.genome.ad.jp/dbget-bin/www_bget?ccr+CC_2068
SIL: http://www.genome.ad.jp/dbget-bin/www_bget?sil+SPO0247(dxs)
SIT: http://www.genome.ad.jp/dbget-bin/www_bget?sit+TM1040_2920
RSP: http://www.genome.ad.jp/dbget-bin/www_bget?rsp+RSP_0254(dxsA) http://www.genome.ad.jp/dbget-bin/www_bget?rsp+RSP_1134(dxs)
JAN: http://www.genome.ad.jp/dbget-bin/www_bget?jan+Jann_0088 http://www.genome.ad.jp/dbget-bin/www_bget?jan+Jann_0170
RDE: http://www.genome.ad.jp/dbget-bin/www_bget?rde+RD1_0101(dxs) http://www.genome.ad.jp/dbget-bin/www_bget?rde+RD1_0548(dxs)
MMR: http://www.genome.ad.jp/dbget-bin/www_bget?mmr+Mmar10_0849
HNE: http://www.genome.ad.jp/dbget-bin/www_bget?hne+HNE_1838(dxs)
ZMO: http://www.genome.ad.jp/dbget-bin/www_bget?zmo+ZMO1234(dxs) http://www.genome.ad.jp/dbget-bin/www_bget?zmo+ZMO1598(dxs)
NAR: http://www.genome.ad.jp/dbget-bin/www_bget?nar+Saro_0161
SAL: http://www.genome.ad.jp/dbget-bin/www_bget?sal+Sala_2354
ELI: http://www.genome.ad.jp/dbget-bin/www_bget?eli+ELI_12520
GOX: http://www.genome.ad.jp/dbget-bin/www_bget?gox+GOX0252
GBE: http://www.genome.ad.jp/dbget-bin/www_bget?gbe+GbCGDNIH1_0221 http://www.genome.ad.jp/dbget-bin/www_bget?gbe+GbCGDNIH1_2404
RRU: http://www.genome.ad.jp/dbget-bin/www_bget?rru+Rru_A0054 http://www.genome.ad.jp/dbget-bin/www_bget?rru+Rru_A2619
MAG: http://www.genome.ad.jp/dbget-bin/www_bget?mag+amb2904
MGM: http://www.genome.ad.jp/dbget-bin/www_bget?mgm+Mmc1_1048
SUS: http://www.genome.ad.jp/dbget-bin/www_bget?sus+Acid_1783
BSU: http://www.genome.ad.jp/dbget-bin/www_bget?bsu+BG11715(dxs)
BHA: http://www.genome.ad.jp/dbget-bin/www_bget?bha+BH2779
BAN: http://www.genome.ad.jp/dbget-bin/www_bget?ban+BA4400(dxs)
BAR: http://www.genome.ad.jp/dbget-bin/www_bget?bar+GBAA4400(dxs)
BAA: http://www.genome.ad.jp/dbget-bin/www_bget?baa+BA_4853
BAT: http://www.genome.ad.jp/dbget-bin/www_bget?bat+BAS4081
BCE: http://www.genome.ad.jp/dbget-bin/www_bget?bce+BC4176(dxs)
BCA: http://www.genome.ad.jp/dbget-bin/www_bget?bca+BCE_4249(dxs)
BCZ: http://www.genome.ad.jp/dbget-bin/www_bget?bcz+BCZK3930(dxs)
BTK: http://www.genome.ad.jp/dbget-bin/www_bget?btk+BT9727_3919(dxs)
BTL: http://www.genome.ad.jp/dbget-bin/www_bget?btl+BALH_3785(dxs)
BLI: http://www.genome.ad.jp/dbget-bin/www_bget?bli+BL01523(dxs)
BLD: http://www.genome.ad.jp/dbget-bin/www_bget?bld+BLi02598(dxs)
BCL: http://www.genome.ad.jp/dbget-bin/www_bget?bcl+ABC2462(dxs)
BAY: http://www.genome.ad.jp/dbget-bin/www_bget?bay+RBAM_022600
BPU: http://www.genome.ad.jp/dbget-bin/www_bget?bpu+BPUM_2159
GKA: http://www.genome.ad.jp/dbget-bin/www_bget?gka+GK2392
GTN: http://www.genome.ad.jp/dbget-bin/www_bget?gtn+GTNG_2322
LMO: http://www.genome.ad.jp/dbget-bin/www_bget?lmo+lmo1365(tktB)
LMF: http://www.genome.ad.jp/dbget-bin/www_bget?lmf+LMOf2365_1382(dxs)
LIN: http://www.genome.ad.jp/dbget-bin/www_bget?lin+lin1402(tktB)
LWE: http://www.genome.ad.jp/dbget-bin/www_bget?lwe+lwe1380(tktB)
LLA: http://www.genome.ad.jp/dbget-bin/www_bget?lla+L108911(dxsA) http://www.genome.ad.jp/dbget-bin/www_bget?lla+L123365(dxsB)
LLC: http://www.genome.ad.jp/dbget-bin/www_bget?llc+LACR_1572 http://www.genome.ad.jp/dbget-bin/www_bget?llc+LACR_1843
LLM: http://www.genome.ad.jp/dbget-bin/www_bget?llm+llmg_0749(dxsB)
SAK: http://www.genome.ad.jp/dbget-bin/www_bget?sak+SAK_0263
LPL: http://www.genome.ad.jp/dbget-bin/www_bget?lpl+lp_2610(dxs)
LJO: http://www.genome.ad.jp/dbget-bin/www_bget?ljo+LJ0406
LAC: http://www.genome.ad.jp/dbget-bin/www_bget?lac+LBA0356
LSL: http://www.genome.ad.jp/dbget-bin/www_bget?lsl+LSL_0209(dxs)
LGA: http://www.genome.ad.jp/dbget-bin/www_bget?lga+LGAS_0350
STH: http://www.genome.ad.jp/dbget-bin/www_bget?sth+STH1842
CAC: http://www.genome.ad.jp/dbget-bin/www_bget?cac+CAC2077 http://www.genome.ad.jp/dbget-bin/www_bget?cac+CA_P0106(dxs)
CPE: http://www.genome.ad.jp/dbget-bin/www_bget?cpe+CPE1819
CPF: http://www.genome.ad.jp/dbget-bin/www_bget?cpf+CPF_2073(dxs)
CPR: http://www.genome.ad.jp/dbget-bin/www_bget?cpr+CPR_1787(dxs)
CTC: http://www.genome.ad.jp/dbget-bin/www_bget?ctc+CTC01575
CNO: http://www.genome.ad.jp/dbget-bin/www_bget?cno+NT01CX_1983
CTH: http://www.genome.ad.jp/dbget-bin/www_bget?cth+Cthe_0828
CDF: http://www.genome.ad.jp/dbget-bin/www_bget?cdf+CD1207(dxs)
CBO: http://www.genome.ad.jp/dbget-bin/www_bget?cbo+CBO1881(dxs)
CBA: http://www.genome.ad.jp/dbget-bin/www_bget?cba+CLB_1818(dxs)
CBH: http://www.genome.ad.jp/dbget-bin/www_bget?cbh+CLC_1825(dxs)
CBF: http://www.genome.ad.jp/dbget-bin/www_bget?cbf+CLI_1945(dxs)
CKL: http://www.genome.ad.jp/dbget-bin/www_bget?ckl+CKL_1231(dxs)
CHY: http://www.genome.ad.jp/dbget-bin/www_bget?chy+CHY_1985(dxs)
DSY: http://www.genome.ad.jp/dbget-bin/www_bget?dsy+DSY2348
DRM: http://www.genome.ad.jp/dbget-bin/www_bget?drm+Dred_1078
PTH: http://www.genome.ad.jp/dbget-bin/www_bget?pth+PTH_1196(dxs)
SWO: http://www.genome.ad.jp/dbget-bin/www_bget?swo+Swol_0582
CSC: http://www.genome.ad.jp/dbget-bin/www_bget?csc+Csac_1853
TTE: http://www.genome.ad.jp/dbget-bin/www_bget?tte+TTE1298(dxs)
MTA: http://www.genome.ad.jp/dbget-bin/www_bget?mta+Moth_1511
MPE: http://www.genome.ad.jp/dbget-bin/www_bget?mpe+MYPE730
MGA: http://www.genome.ad.jp/dbget-bin/www_bget?mga+MGA_1268(dxs)
MTU: http://www.genome.ad.jp/dbget-bin/www_bget?mtu+Rv2682c(dxs1) http://www.genome.ad.jp/dbget-bin/www_bget?mtu+Rv3379c(dxs2)
MTC: http://www.genome.ad.jp/dbget-bin/www_bget?mtc+MT2756(dxs)
MBO: http://www.genome.ad.jp/dbget-bin/www_bget?mbo+Mb2701c(dxs1) http://www.genome.ad.jp/dbget-bin/www_bget?mbo+Mb3413c(dxs2)
MLE: http://www.genome.ad.jp/dbget-bin/www_bget?mle+ML1038(dxs)
MPA: http://www.genome.ad.jp/dbget-bin/www_bget?mpa+MAP2803c(dxs)
MAV: http://www.genome.ad.jp/dbget-bin/www_bget?mav+MAV_3577(dxs)
MSM: http://www.genome.ad.jp/dbget-bin/www_bget?msm+MSMEG_2776(dxs)
MMC: http://www.genome.ad.jp/dbget-bin/www_bget?mmc+Mmcs_2208
CGL: http://www.genome.ad.jp/dbget-bin/www_bget?cgl+NCgl1827(cgl1902)
CGB: http://www.genome.ad.jp/dbget-bin/www_bget?cgb+cg2083(dxs)
CEF: http://www.genome.ad.jp/dbget-bin/www_bget?cef+CE1796
CDI: http://www.genome.ad.jp/dbget-bin/www_bget?cdi+DIP1397(dxs)
CJK: http://www.genome.ad.jp/dbget-bin/www_bget?cjk+jk1078(dxs)
NFA: http://www.genome.ad.jp/dbget-bin/www_bget?nfa+nfa37410(dxs)
RHA: http://www.genome.ad.jp/dbget-bin/www_bget?rha+RHA1_ro06843
SCO: http://www.genome.ad.jp/dbget-bin/www_bget?sco+SCO6013(SC1C3.01) http://www.genome.ad.jp/dbget-bin/www_bget?sco+SCO6768(SC6A5.17)
SMA: http://www.genome.ad.jp/dbget-bin/www_bget?sma+SAV1646(dxs1) http://www.genome.ad.jp/dbget-bin/www_bget?sma+SAV2244(dxs2)
TWH: http://www.genome.ad.jp/dbget-bin/www_bget?twh+TWT484
TWS: http://www.genome.ad.jp/dbget-bin/www_bget?tws+TW280(Dxs)
LXX: http://www.genome.ad.jp/dbget-bin/www_bget?lxx+Lxx10450(dxs)
CMI: http://www.genome.ad.jp/dbget-bin/www_bget?cmi+CMM_1660(dxsA)
AAU: http://www.genome.ad.jp/dbget-bin/www_bget?aau+AAur_1790(dxs)
PAC: http://www.genome.ad.jp/dbget-bin/www_bget?pac+PPA1062
TFU: http://www.genome.ad.jp/dbget-bin/www_bget?tfu+Tfu_1917
FRA: http://www.genome.ad.jp/dbget-bin/www_bget?fra+Francci3_1326
FAL: http://www.genome.ad.jp/dbget-bin/www_bget?fal+FRAAL2088(dxs)
ACE: http://www.genome.ad.jp/dbget-bin/www_bget?ace+Acel_1393
SEN: http://www.genome.ad.jp/dbget-bin/www_bget?sen+SACE_1815(dxs) http://www.genome.ad.jp/dbget-bin/www_bget?sen+SACE_4351
BLO: http://www.genome.ad.jp/dbget-bin/www_bget?blo+BL1132(dxs)
BAD: http://www.genome.ad.jp/dbget-bin/www_bget?bad+BAD_0513(dxs)
FNU: http://www.genome.ad.jp/dbget-bin/www_bget?fnu+FN1208 http://www.genome.ad.jp/dbget-bin/www_bget?fnu+FN1464
RBA: http://www.genome.ad.jp/dbget-bin/www_bget?rba+RB2143(dxs)
CTR: http://www.genome.ad.jp/dbget-bin/www_bget?ctr+CT331(dxs)
CTA: http://www.genome.ad.jp/dbget-bin/www_bget?cta+CTA_0359(dxs)
CMU: http://www.genome.ad.jp/dbget-bin/www_bget?cmu+TC0608
CPN: http://www.genome.ad.jp/dbget-bin/www_bget?cpn+CPn1060(tktB_2)
CPA: http://www.genome.ad.jp/dbget-bin/www_bget?cpa+CP0790
CPJ: http://www.genome.ad.jp/dbget-bin/www_bget?cpj+CPj1060(tktB_2)
CPT: http://www.genome.ad.jp/dbget-bin/www_bget?cpt+CpB1102
CCA: http://www.genome.ad.jp/dbget-bin/www_bget?cca+CCA00304(dxs)
CAB: http://www.genome.ad.jp/dbget-bin/www_bget?cab+CAB301(dxs)
CFE: http://www.genome.ad.jp/dbget-bin/www_bget?cfe+CF0699(dxs)
PCU: http://www.genome.ad.jp/dbget-bin/www_bget?pcu+pc0619(dxs)
TPA: http://www.genome.ad.jp/dbget-bin/www_bget?tpa+TP0824
TDE: http://www.genome.ad.jp/dbget-bin/www_bget?tde+TDE1910(dxs)
LIL: http://www.genome.ad.jp/dbget-bin/www_bget?lil+LA3285(dxs)
LIC: http://www.genome.ad.jp/dbget-bin/www_bget?lic+LIC10863(dxs)
LBJ: http://www.genome.ad.jp/dbget-bin/www_bget?lbj+LBJ_0917(dxs)
LBL: http://www.genome.ad.jp/dbget-bin/www_bget?lbl+LBL_0932(dxs)
SYN: http://www.genome.ad.jp/dbget-bin/www_bget?syn+sll1945(dxs)
SYW: http://www.genome.ad.jp/dbget-bin/www_bget?syw+SYNW1292(Dxs)
SYC: http://www.genome.ad.jp/dbget-bin/www_bget?syc+syc1087_c(dxs)
SYF: http://www.genome.ad.jp/dbget-bin/www_bget?syf+Synpcc7942_0430
SYD: http://www.genome.ad.jp/dbget-bin/www_bget?syd+Syncc9605_1430
SYE: http://www.genome.ad.jp/dbget-bin/www_bget?sye+Syncc9902_1069
SYG: http://www.genome.ad.jp/dbget-bin/www_bget?syg+sync_1410(dxs)
SYR: http://www.genome.ad.jp/dbget-bin/www_bget?syr+SynRCC307_1390(dxs)
SYX: http://www.genome.ad.jp/dbget-bin/www_bget?syx+SynWH7803_1223(dxs)
CYA: http://www.genome.ad.jp/dbget-bin/www_bget?cya+CYA_1701(dxs)
CYB: http://www.genome.ad.jp/dbget-bin/www_bget?cyb+CYB_1983(dxs)
TEL: http://www.genome.ad.jp/dbget-bin/www_bget?tel+tll0623
GVI: http://www.genome.ad.jp/dbget-bin/www_bget?gvi+gll0194
ANA: http://www.genome.ad.jp/dbget-bin/www_bget?ana+alr0599
AVA: http://www.genome.ad.jp/dbget-bin/www_bget?ava+Ava_4532
PMA: http://www.genome.ad.jp/dbget-bin/www_bget?pma+Pro0928(dxs)
PMM: http://www.genome.ad.jp/dbget-bin/www_bget?pmm+PMM0907(dxs)
PMT: http://www.genome.ad.jp/dbget-bin/www_bget?pmt+PMT0685(dxs)
PMN: http://www.genome.ad.jp/dbget-bin/www_bget?pmn+PMN2A_0300
PMI: http://www.genome.ad.jp/dbget-bin/www_bget?pmi+PMT9312_0893
PMB: http://www.genome.ad.jp/dbget-bin/www_bget?pmb+A9601_09541(dxs)
PMC: http://www.genome.ad.jp/dbget-bin/www_bget?pmc+P9515_09901(dxs)
PMF: http://www.genome.ad.jp/dbget-bin/www_bget?pmf+P9303_15371(dxs)
PMG: http://www.genome.ad.jp/dbget-bin/www_bget?pmg+P9301_09521(dxs)
PMH: http://www.genome.ad.jp/dbget-bin/www_bget?pmh+P9215_09851
PMJ: http://www.genome.ad.jp/dbget-bin/www_bget?pmj+P9211_08521
PME: http://www.genome.ad.jp/dbget-bin/www_bget?pme+NATL1_09721(dxs)
TER: http://www.genome.ad.jp/dbget-bin/www_bget?ter+Tery_3042
BTH: http://www.genome.ad.jp/dbget-bin/www_bget?bth+BT_1403 http://www.genome.ad.jp/dbget-bin/www_bget?bth+BT_4099
BFR: http://www.genome.ad.jp/dbget-bin/www_bget?bfr+BF0873 http://www.genome.ad.jp/dbget-bin/www_bget?bfr+BF4306
BFS: http://www.genome.ad.jp/dbget-bin/www_bget?bfs+BF0796(dxs) http://www.genome.ad.jp/dbget-bin/www_bget?bfs+BF4114
PGI: http://www.genome.ad.jp/dbget-bin/www_bget?pgi+PG2217(dxs)
CHU: http://www.genome.ad.jp/dbget-bin/www_bget?chu+CHU_3643(dxs)
GFO: http://www.genome.ad.jp/dbget-bin/www_bget?gfo+GFO_3470(dxs)
FPS: http://www.genome.ad.jp/dbget-bin/www_bget?fps+FP0279(dxs)
CTE: http://www.genome.ad.jp/dbget-bin/www_bget?cte+CT0337(dxs)
CPH: http://www.genome.ad.jp/dbget-bin/www_bget?cph+Cpha266_0671
PVI: http://www.genome.ad.jp/dbget-bin/www_bget?pvi+Cvib_0498
PLT: http://www.genome.ad.jp/dbget-bin/www_bget?plt+Plut_0450
DET: http://www.genome.ad.jp/dbget-bin/www_bget?det+DET0745(dxs)
DEH: http://www.genome.ad.jp/dbget-bin/www_bget?deh+cbdb_A720(dxs)
DRA: http://www.genome.ad.jp/dbget-bin/www_bget?dra+DR_1475
DGE: http://www.genome.ad.jp/dbget-bin/www_bget?dge+Dgeo_0994
TTH: http://www.genome.ad.jp/dbget-bin/www_bget?tth+TTC1614
TTJ: http://www.genome.ad.jp/dbget-bin/www_bget?ttj+TTHA0006
AAE: http://www.genome.ad.jp/dbget-bin/www_bget?aae+aq_881
TMA: http://www.genome.ad.jp/dbget-bin/www_bget?tma+TM1770
PMO: http://www.genome.ad.jp/dbget-bin/www_bget?pmo+Pmob_1001
Репрезентативные нуклеиновые кислоты и полипептиды ацетил-СоА-ацетилтрансферазы:
HSA: 38(ACAT1) 39(ACAT2)
PTR: 451528(ACAT1)
MCC: 707653(ACAT1) 708750(ACAT2)
MMU: 110446(Acat1) 110460(Acat2)
RNO: 25014(Acat1)
CFA: 484063(ACAT2) 489421(ACAT1)
GGA: 418968(ACAT1) 421587(RCJMB04_34i5)
XLA: 379569(MGC69098) 414622(MGC81403) 414639(MGC81256) 444457(MGC83664)
XTR: 394562(acat2)
DRE: 30643(acat2)
SPU: 759502(LOC759502)
DME: Dmel_CG10932 Dmel_CG9149
CEL: T02G5.4 T02G5.7 T02G5.8(kat-1)
ATH: AT5G48230(ACAT2/EMB1276)
OSA: 4326136 4346520
CME: CMA042C CME087C
SCE: YPL028W(ERG10)
AGO: AGOS_ADR165C
PIC: PICST_31707(ERG10)
CAL: CaO19.1591(erg10)
CGR: CAGL0L12364g
SPO: SPBC215.09c
MGR: MGG_01755 MGG_13499
ANI: AN1409.2
AFM: AFUA_6G14200 AFUA_8G04000
AOR: AO090103000012 AO090103000406
CNE: CNC05280
UMA: UM03571.1
DDI: DDB_0231621
PFA: PF14_0484
TET: TTHERM_00091590 TTHERM_00277470 TTHERM_00926980
TCR: 511003.60
ECO: b2224(atoB)
ECJ: JW2218(atoB) JW5453(yqeF)
ECE: Z4164(yqeF)
ECS: ECs3701
ECC: c2767(atoB) c3441(yqeF)
ECI: UTI89_C2506(atoB) UTI89_C3247(yqeF)
ECP: ECP_2268 ECP_2857
ECV: APECO1_3662(yqeF) APECO1_4335(atoB) APECO1_43352(atoB)
ECX: EcHS_A2365
STY: STY3164(yqeF)
STT: t2929(yqeF)
SPT: SPA2886(yqeF)
SEC: SC2958(yqeF)
STM: STM3019(yqeF)
SFL: SF2854(yqeF)
SFX: S3052(yqeF)
SFV: SFV_2922(yqeF)
SSN: SSON_2283(atoB) SSON_3004(yqeF)
SBO: SBO_2736(yqeF)
ECA: ECA1282(atoB)
ENT: Ent638_3299
SPE: Spro_0592
HIT: NTHI0932(atoB)
XCC: XCC1297(atoB)
XCB: XC_2943
XCV: XCV1401(thlA)
XAC: XAC1348(atoB)
XOO: XOO1881(atoB)
XOM: XOO_1778(XOO1778)
VCH: VCA0690
VCO: VC0395_0630
VVU: VV2_0494 VV2_0741
VVY: VVA1043 VVA1210
VPA: VPA0620 VPA1123 VPA1204
PPR: PBPRB1112 PBPRB1840
PAE: PA2001(atoB) PA2553 PA3454 PA3589 PA3925
PAU: PA14_38630(atoB)
PPU: PP_2051(atoB) PP_2215(fadAx) PP_3754 PP_4636
PPF: Pput_2009 Pput_2403 Pput_3523 Pput_4498
PST: PSPTO_0957(phbA-1) PSPTO_3164(phbA-2)
PSB: Psyr_0824 Psyr_3031
PSP: PSPPH_0850(phbA1) PSPPH_2209(phbA2)
PFL: PFL_1478(atoB-2) PFL_2321 PFL_3066 PFL_4330(atoB-2) PFL_5283
PFO: Pfl_1269 Pfl_1739 Pfl_2074 Pfl_2868
PEN: PSEEN3197 PSEEN3547(fadAx) PSEEN4635(phbA)
PMY: Pmen_1138 Pmen_2036 Pmen_3597 Pmen_3662 Pmen_3820
PAR: Psyc_0252 Psyc_1169
PCR: Pcryo_0278 Pcryo_1236 Pcryo_1260
PRW: PsycPRwf_2011
ACI: ACIAD0694 ACIAD1612 ACIAD2516(atoB)
SON: SO_1677(atoB)
SDN: Sden_1943
SFR: Sfri_1338 Sfri_2063
SAZ: Sama_1375
SBL: Sbal_1495
SBM: Shew185_1489
SBN: Sbal195_1525
SLO: Shew_1667 Shew_2858
SPC: Sputcn32_1397
SSE: Ssed_1473 Ssed_3533
SPL: Spea_2783
SHE: Shewmr4_2597
SHM: Shewmr7_2664
SHN: Shewana3_2771
SHW: Sputw3181_2704
ILO: IL0872
CPS: CPS_1605 CPS_2626
PHA: PSHAa0908 PSHAa1454(atoB) PSHAa1586(atoB)
PAT: Patl_2923
SDE: Sde_3149
PIN: Ping_0659 Ping_2401
MAQ: Maqu_2117 Maqu_2489 Maqu_2696 Maqu_3162
CBU: CBU_0974
LPN: lpg1825(atoB)
LPF: lpl1789
LPP: lpp1788
NOC: Noc_1891
AEH: Mlg_0688 Mlg_2706
HHA: Hhal_1685
HCH: HCH_05299
CSA: Csal_0301 Csal_3068
ABO: ABO_0648(fadAx)
MMW: Mmwyl1_0073 Mmwyl1_3021 Mmwyl1_3053 Mmwyl1_3097 Mmwyl1_4182
AHA: AHA_2143(atoB)
CVI: CV_2088(atoB) CV_2790(phaA)
RSO: RSc0276(atoB) RSc1632(phbA) RSc1637(bktB) RSc1761(RS02948)
REU: Reut_A0138 Reut_A1348 Reut_A1353 Reut_B4561 Reut_B4738 Reut_B5587 Reut_C5943 Reut_C6062
REH: H16_A0170 H16_A0867 H16_A0868 H16_A0872 H16_A1297 H16_A1438(phaA) H16_A1445(bktB) H16_A1528 H16_A1713 H16_A1720 H16_A1887 H16_A2148 H16_B0380 H16_B0381 H16_B0406 H16_B0662 H16_B0668 H16_B0759 H16_B1369 H16_B1771
RME: Rmet_0106 Rmet_1357 Rmet_1362 Rmet_5156
BMA: BMA1316 BMA1321(phbA) BMA1436
BMV: BMASAVP1_A1805(bktB) BMASAVP1_A1810(phbA)
BML: BMA10299_A0086(phbA) BMA10299_A0091
BMN: BMA10247_1076(bktB) BMA10247_1081(phbA)
BXE: Bxe_A2273 Bxe_A2335 Bxe_A2342 Bxe_A4255 Bxe_B0377 Bxe_B0739 Bxe_C0332 Bxe_C0574 Bxe_C0915
BVI: Bcep1808_0519 Bcep1808_1717 Bcep1808_2877 Bcep1808_3594 Bcep1808_4015 Bcep1808_5507 Bcep1808_5644
BUR: Bcep18194_A3629 Bcep18194_A5080 Bcep18194_A5091 Bcep18194_A6102 Bcep18194_B0263 Bcep18194_B1439 Bcep18194_C6652 Bcep18194_C6802 Bcep18194_C6874 Bcep18194_C7118 Bcep18194_C7151 Bcep18194_C7332
BCN: Bcen_1553 Bcen_1599 Bcen_2158 Bcen_2563 Bcen_2998 Bcen_6289
BCH: Bcen2424_0542 Bcen2424_1790 Bcen2424_2772 Bcen2424_5368 Bcen2424_6232 Bcen2424_6276
BAM: Bamb_0447 Bamb_1728 Bamb_2824 Bamb_4717 Bamb_5771 Bamb_5969
BPS: BPSL1426 BPSL1535(phbA) BPSL1540
BPM: BURPS1710b_2325(bktB) BURPS1710b_2330(phbA) BURPS1710b_2453(atoB-2)
BPL: BURPS1106A_2197(bktB) BURPS1106A_2202(phbA)
BPD: BURPS668_2160(bktB) BURPS668_2165(phbA)
BTE: BTH_I2144 BTH_I2256 BTH_I2261
PNU: Pnuc_0927
BPE: BP0447 BP0668 BP2059
BPA: BPP0608 BPP1744 BPP3805 BPP4216 BPP4361
BBR: BB0614 BB3364 BB4250 BB4804 BB4947
RFR: Rfer_0272 Rfer_1000 Rfer_1871 Rfer_2273 Rfer_2561 Rfer_2594 Rfer_3839
POL: Bpro_1577 Bpro_2140 Bpro_3113 Bpro_4187
PNA: Pnap_0060 Pnap_0458 Pnap_0867 Pnap_1159 Pnap_2136 Pnap_2804
AAV: Aave_0031 Aave_2478 Aave_3944 Aave_4368
AJS: Ajs_0014 Ajs_0124 Ajs_1931 Ajs_2073 Ajs_2317 Ajs_3548 Ajs_3738 Ajs_3776
VEI: Veis_1331 Veis_3818 Veis_4193
DAC: Daci_0025 Daci_0192 Daci_3601 Daci_5988
MPT: Mpe_A1536 Mpe_A1776 Mpe_A1869 Mpe_A3367
HAR: HEAR0577(phbA)
MMS: mma_0555
NEU: NE2262(bktB)
NET: Neut_0610
EBA: ebA5202 p2A409(tioL)
AZO: azo0464(fadA1) azo0469(fadA2) azo2172(thlA)
DAR: Daro_0098 Daro_3022
HPA: HPAG1_0675
HAC: Hac_0958(atoB)
GME: Gmet_1719 Gmet_2074 Gmet_2213 Gmet_2268 Gmet_3302
GUR: Gura_3043
BBA: Bd0404(atoB) Bd2095
DOL: Dole_0671 Dole_1778 Dole_2160 Dole_2187
ADE: Adeh_0062 Adeh_2365
AFW: Anae109_0064 Anae109_1504
MXA: MXAN_3791
SAT: SYN_02642
SFU: Sfum_2280 Sfum_3582
RPR: RP737
RCO: RC1134 RC1135
RFE: RF_0163(paaJ)
RBE: RBE_0139(paaJ)
RAK: A1C_05820
RBO: A1I_07215
RCM: A1E_04760
PUB: SAR11_0428(thlA)
MLO: mlr3847
MES: Meso_3374
PLA: Plav_1573 Plav_2783
SME: SMa1450 SMc03879(phbA)
SMD: Smed_0499 Smed_3117 Smed_5094 Smed_5096
ATU: Atu2769(atoB) Atu3475
ATC: AGR_C_5022(phbA) AGR_L_2713
RET: RHE_CH04018(phbAch) RHE_PC00068(ypc00040) RHE_PF00014(phbAf)
RLE: RL4621(phaA) pRL100301 pRL120369
BME: BMEI0274 BMEII0817
BMF: BAB1_1783(phbA-1) BAB2_0790(phbA-2)
BMS: BR1772(phbA-1) BRA0448(phbA-2)
BMB: BruAb1_1756(phbA-1) BruAb2_0774(phbA-2)
BOV: BOV_1707(phbA-1)
OAN: Oant_1130 Oant_3107 Oant_3718 Oant_4020
BJA: bll0226(atoB) bll3949 bll7400 bll7819 blr3724(phbA)
BRA: BRADO0562(phbA) BRADO0983(pimB) BRADO3110 BRADO3134(atoB)
BBT: BBta_3558 BBta_3575(atoB) BBta_5147(pimB) BBta_7072(pimB) BBta_7614(phbA)
RPA: RPA0513(pcaF) RPA0531 RPA3715(pimB)
RPB: RPB_0509 RPB_0525 RPB_1748
RPC: RPC_0504 RPC_0636 RPC_0641 RPC_0832 RPC_1050 RPC_2005 RPC_2194 RPC_2228
RPD: RPD_0306 RPD_0320 RPD_3105 RPD_3306
RPE: RPE_0168 RPE_0248 RPE_3827
NWI: Nwi_3060
XAU: Xaut_3108 Xaut_4665
CCR: CC_0510 CC_0894 CC_3462
SIL: SPO0142(bktB) SPO0326(phbA) SPO0773 SPO3408
SIT: TM1040_0067 TM1040_2790 TM1040_3026 TM1040_3735
RSP: RSP_0745 RSP_1354 RSP_3184
RSH: Rsph17029_0022 Rsph17029_2401 Rsph17029_3179 Rsph17029_3921
RSQ: Rsph17025_0012 Rsph17025_2466 Rsph17025_2833
JAN: Jann_0262 Jann_0493 Jann_4050
RDE: RD1_0025 RD1_0201(bktB) RD1_3394(phbA)
PDE: Pden_2026 Pden_2663 Pden_2870 Pden_2907 Pden_4811 Pden_5022
DSH: Dshi_0074 Dshi_3066 Dshi_3331
MMR: Mmar10_0697
HNE: HNE_2706 HNE_3065 HNE_3133
NAR: Saro_0809 Saro_1069 Saro_1222 Saro_2306 Saro_2349
SAL: Sala_0781 Sala_1244 Sala_2896 Sala_3158
SWI: Swit_0632 Swit_0752 Swit_2893 Swit_3602 Swit_4887 Swit_5019 Swit_5309
ELI: ELI_01475 ELI_06705 ELI_12035
GBE: GbCGDNIH1_0447
ACR: Acry_1847 Acry_2256
RRU: Rru_A0274 Rru_A1380 Rru_A1469 Rru_A1946 Rru_A3387
MAG: amb0842
MGM: Mmc1_1165
ABA: Acid345_3239
BSU: BG11319(mmgA) BG13063(yhfS)
BHA: BH1997 BH2029 BH3801(mmgA)
BAN: BA3687 BA4240 BA5589
BAR: GBAA3687 GBAA4240 GBAA5589
BAA: BA_0445 BA_4172 BA_4700
BAT: BAS3418 BAS3932 BAS5193
BCE: BC3627 BC4023 BC5344
BCA: BCE_3646 BCE_4076 BCE_5475
BCZ: BCZK3329(mmgA) BCZK3780(thl) BCZK5044(atoB)
BCY: Bcer98_2722 Bcer98_3865
BTK: BT9727_3379(mmgA) BT9727_3765(thl) BT9727_5028(atoB)
BTL: BALH_3262(mmgA) BALH_3642(fadA) BALH_4843(atoB)
BLI: BL03925(mmgA)
BLD: BLi03968(mmgA)
BCL: ABC0345 ABC2989 ABC3617 ABC3891(mmgA)
BAY: RBAM_022450
BPU: BPUM_2374(yhfS) BPUM_2941 BPUM_3373
OIH: OB0676 OB0689 OB2632 OB3013
GKA: GK1658 GK3397
SAU: SA0342 SA0534(vraB)
SAV: SAV0354 SAV0576(vraB)
SAM: MW0330 MW0531(vraB)
SAR: SAR0351(thl) SAR0581
SAS: SAS0330 SAS0534
SAC: SACOL0426 SACOL0622(atoB)
SAB: SAB0304(th1) SAB0526
SAA: SAUSA300_0355 SAUSA300_0560(vraB)
SAO: SAOUHSC_00336 SAOUHSC_00558
SAJ: SaurJH9_0402
SAH: SaurJH1_0412
SEP: SE0346 SE2384
SER: SERP0032 SERP0220
SHA: SH0510(mvaC) SH2417
SSP: SSP0325 SSP2145
LMO: lmo1414
LMF: LMOf2365_1433
LIN: lin1453
LWE: lwe1431
LLA: L11745(thiL) L25946(fadA)
LLC: LACR_1665 LACR_1956
LLM: llmg_0930(thiL)
SPY: SPy_0140 SPy_1637(atoB)
SPZ: M5005_Spy_0119 M5005_Spy_0432 M5005_Spy_1344(atoB)
SPM: spyM18_0136 spyM18_1645(atoB)
SPG: SpyM3_0108 SpyM3_1378(atoB)
SPS: SPs0110 SPs0484
SPH: MGAS10270_Spy0121 MGAS10270_Spy0433 MGAS10270_Spy1461(atoB)
SPI: MGAS10750_Spy0124 MGAS10750_Spy0452 MGAS10750_Spy1453(atoB)
SPJ: MGAS2096_Spy0123 MGAS2096_Spy0451 MGAS2096_Spy1365(atoB)
SPK: MGAS9429_Spy0121 MGAS9429_Spy0431 MGAS9429_Spy1339(atoB)
SPF: SpyM50447(atoB2)
SPA: M6_Spy0166 M6_Spy0466 M6_Spy1390
SPB: M28_Spy0117 M28_Spy0420 M28_Spy1385(atoB)
SAK: SAK_0568
LJO: LJ1609
LAC: LBA0626(thiL)
LSA: LSA1486
LDB: Ldb0879
LBU: LBUL_0804
LBR: LVIS_2218
LCA: LSEI_1787
LGA: LGAS_1374
LRE: Lreu_0052
EFA: EF1364
OOE: OEOE_0529
STH: STH2913 STH725 STH804
CAC: CAC2873 CA_P0078(thiL)
CPE: CPE2195(atoB)
CPF: CPF_2460
CPR: CPR_2170
CTC: CTC00312
CNO: NT01CX_0538 NT01CX_0603
CDF: CD1059(thlA1) CD2676(thlA2)
CBO: CBO3200(thl)
CBE: Cbei_0411 Cbei_3630
CKL: CKL_3696(thlA1) CKL_3697(thlA2) CKL_3698(thlA3)
AMT: Amet_4630
AOE: Clos_0084 Clos_0258
CHY: CHY_1288 CHY_1355(atoB) CHY_1604 CHY_1738
DSY: DSY0632 DSY0639 DSY1567 DSY1710 DSY2402 DSY3302
DRM: Dred_0400 Dred_1491 Dred_1784 Dred_1892
SWO: Swol_0308 Swol_0675 Swol_0789 Swol_1486 Swol_1934 Swol_2051
TTE: TTE0549(paaJ)
MTA: Moth_1260
MTU: Rv1135A Rv1323(fadA4) Rv3546(fadA5)
MTC: MT1365(phbA)
MBO: Mb1167 Mb1358(fadA4) Mb3576(fadA5) Mb3586c(fadA6)
MBB: BCG_1197 BCG_1385(fadA4) BCG_3610(fadA5) BCG_3620c(fadA6)
MLE: ML1158(fadA4)
MPA: MAP2407c(fadA3) MAP2436c(fadA4)
MAV: MAV_1544 MAV_1573 MAV_1863 MAV_5081
MSM: MSMEG_2224 MSMEG_4920
MUL: MUL_0357
MVA: Mvan_1976 Mvan_1988 Mvan_4305 Mvan_4677 Mvan_4891
MGI: Mflv_1347 Mflv_1484 Mflv_2040 Mflv_2340 Mflv_4356 Mflv_4368
MMC: Mmcs_1758 Mmcs_1769 Mmcs_3796 Mmcs_3864
MKM: Mkms_0251 Mkms_1540 Mkms_1805 Mkms_1816 Mkms_2836 Mkms_3159 Mkms_3286 Mkms_3869 Mkms_3938 Mkms_4227 Mkms_4411 Mkms_4580 Mkms_4724 Mkms_4764 Mkms_4776
MJL: Mjls_0231 Mjls_1739 Mjls_1750 Mjls_2819 Mjls_3119 Mjls_3235 Mjls_3800 Mjls_3850 Mjls_4110 Mjls_4383 Mjls_4705 Mjls_4876 Mjls_5018 Mjls_5063 Mjls_5075
CGL: NCgl2309(cgl2392)
CGB: cg2625(pcaF)
CEF: CE0731 CE2295
CJK: jk1543(fadA3)
NFA: nfa10750(fadA4)
RHA: RHA1_ro01455 RHA1_ro01623 RHA1_ro01876 RHA1_ro02517(catF) RHA1_ro03022 RHA1_ro03024 RHA1_ro03391 RHA1_ro03892 RHA1_ro04599 RHA1_ro05257 RHA1_ro08871
SCO: SCO5399(SC8F4.03)
SMA: SAV1384(fadA5) SAV2856(fadA1)
ART: Arth_1160 Arth_2986 Arth_3268 Arth_4073
NCA: Noca_1371 Noca_1797 Noca_1828 Noca_2764 Noca_4142
TFU: Tfu_1520 Tfu_2394
FRA: Francci3_3687
FRE: Franean1_1044 Franean1_2711 Franean1_2726 Franean1_3929 Franean1_4037 Franean1_4577
FAL: FRAAL2514 FRAAL2618 FRAAL5910(atoB)
ACE: Acel_0626 Acel_0672
SEN: SACE_1192(mmgA) SACE_2736(fadA6) SACE_4011(catF) SACE_6236(fadA4)
STP: Strop_3610
SAQ: Sare_1316 Sare_3991
RXY: Rxyl_1582 Rxyl_1842 Rxyl_2389 Rxyl_2530
FNU: FN0495
BGA: BG0110(fadA)
BAF: BAPKO_0110(fadA)
LIL: LA0457(thiL1) LA0828(thiL2) LA4139(fadA)
LIC: LIC10396(phbA)
LBJ: LBJ_2862(paaJ-4)
LBL: LBL_0209(paaJ-4)
SYN: slr1993(phaA)
SRU: SRU_1211(atoB) SRU_1547
CHU: CHU_1910(atoB)
GFO: GFO_1507(atoB)
FJO: Fjoh_4612
FPS: FP0770 FP1586 FP1725
RRS: RoseRS_3911 RoseRS_4348
RCA: Rcas_0702 Rcas_3206
HAU: Haur_0522
DRA: DR_1072 DR_1428 DR_1960 DR_2480 DR_A0053
DGE: Dgeo_0755 Dgeo_1305 Dgeo_1441 Dgeo_1883
TTH: TTC0191 TTC0330
TTJ: TTHA0559
TME: Tmel_1134
FNO: Fnod_0314
PMO: Pmob_0515
HMA: rrnAC0896(acaB3) rrnAC2815(aca2) rrnAC3497(yqeF) rrnB0240(aca1) rrnB0242(acaB2) rrnB0309(acaB1)
TAC: Ta0582
TVO: TVN0649
PTO: PTO1505
APE: APE_2108
SSO: SSO2377(acaB-4)
STO: ST0514
SAI: Saci_0963 Saci_1361(acaB1)
MSE: Msed_0656
PAI: PAE1220
PIS: Pisl_0029 Pisl_1301
PCL: Pcal_0781
PAS: Pars_0309 Pars_1071
CMA: Cmaq_1941
Репрезентативные нуклеиновые кислоты и полипептиды HMG-CoA-синтазы
HSA: 3157(HMGCS1) 3158(HMGCS2)
PTR: 457169(HMGCS2) 461892(HMGCS1)
MCC: 702553(HMGCS1) 713541(HMGCS2)
MMU: 15360(Hmgcs2) 208715(Hmgcs1)
RNO: 24450(Hmgcs2) 29637(Hmgcs1)
CFA: 479344(HMGCS1) 607923(HMGCS2)
BTA: 407767(HMGCS1)
SSC: 397673(CH242-38B5.1)
GGA: 396379(HMGCS1)
XLA: 380091(hmgcs1) 447204(MGC80816)
DRE: 394060(hmgcs1)
SPU: 578259(LOC578259)
DME: Dmel_CG4311(Hmgs)
CEL: F25B4.6
ATH: AT4G11820(BAP1)
OSA: 4331418 4347614
CME: CMM189C
SCE: YML126C(ERG13)
AGO: AGOS_ADL356C
PIC: PICST_83020
CAL: CaO19_7312(CaO19.7312)
CGR: CAGL0H04081g
SPO: SPAC4F8.14c(hcs)
MGR: MGG_01026
ANI: AN4923.2
AFM: AFUA_3G10660 AFUA_8G07210
AOR: AO090003000611 AO090010000487
CNE: CNC05080 CNG02670
UMA: UM05362.1
ECU: ECU10_0510
DDI: DDBDRAFT_0217522 DDB_0219924(hgsA)
TET: TTHERM_00691190
TBR: Tb927.8.6110
YPE: YPO1457
YPK: y2712(pksG)
YPM: YP_1349(pksG)
YPA: YPA_0750
YPN: YPN_2521
YPP: YPDSF_1517
YPS: YPTB1475
CBD: COXBU7E912_1931
TCX: Tcr_1719
DNO: DNO_0799
BMA: BMAA1212
BPS: BPSS1002
BPM: BURPS1710b_A2613
BPL: BURPS1106A_A1384
BPD: BURPS668_A1470
BTE: BTH_II1670
MXA: MXAN_3948(tac) MXAN_4267(mvaS)
BSU: BG10926(pksG)
OIH: OB2248
SAU: SA2334(mvaS)
SAV: SAV2546(mvaS)
SAM: MW2467(mvaS)
SAR: SAR2626(mvaS)
SAS: SAS2432
SAC: SACOL2561
SAB: SAB2420(mvaS)
SAA: SAUSA300_2484
SAO: SAOUHSC_02860
SAJ: SaurJH9_2569
SAH: SaurJH1_2622
SEP: SE2110
SER: SERP2122
SHA: SH0508(mvaS)
SSP: SSP0324
LMO: lmo1415
LMF: LMOf2365_1434(mvaS)
LIN: lin1454
LWE: lwe1432(mvaS)
LLA: L13187(hmcM)
LLC: LACR_1666
LLM: llmg_0929(hmcM)
SPY: SPy_0881(mvaS.2)
SPZ: M5005_Spy_0687(mvaS.1)
SPM: spyM18_0942(mvaS2)
SPG: SpyM3_0600(mvaS.2)
SPS: SPs1253
SPH: MGAS10270_Spy0745(mvaS1)
SPI: MGAS10750_Spy0779(mvaS1)
SPJ: MGAS2096_Spy0759(mvaS1)
SPK: MGAS9429_Spy0743(mvaS1)
SPF: SpyM51121(mvaS)
SPA: M6_Spy0704
SPB: M28_Spy0667(mvaS.1)
SPN: SP_1727
SPR: spr1571(mvaS)
SPD: SPD_1537(mvaS)
SAG: SAG1316
SAN: gbs1386
SAK: SAK_1347
SMU: SMU.943c
STC: str0577(mvaS)
STL: stu0577(mvaS)
STE: STER_0621
SSA: SSA_0338(mvaS)
SSU: SSU05_1641
SSV: SSU98_1652
SGO: SGO_0244
LPL: lp_2067(mvaS)
LJO: LJ1607
LAC: LBA0628(hmcS)
LSA: LSA1484(mvaS)
LSL: LSL_0526
LDB: Ldb0881(mvaS)
LBU: LBUL_0806
LBR: LVIS_1363
LCA: LSEI_1785
LGA: LGAS_1372
LRE: Lreu_0676
PPE: PEPE_0868
EFA: EF1363
OOE: OEOE_0968
LME: LEUM_1184
NFA: nfa22120
SEN: SACE_4570(pksG)
BBU: BB0683
BGA: BG0706
BAF: BAPKO_0727
FJO: Fjoh_0678
HAL: VNG1615G(mvaB)
HMA: rrnAC1740(mvaS)
HWA: HQ2868A(mvaB)
NPH: NP2608A(mvaB_1) NP4836A(mvaB_2)
Репрезентативные нуклеиновые кислоты и полипептиды гидроксиметилглутарил-CoA-редуктазы
HSA: http://www.genome.ad.jp/dbget-bin/www_bget?hsa+3156(HMGCR)
PTR: http://www.genome.ad.jp/dbget-bin/www_bget?ptr+471516(HMGCR)
MCC: http://www.genome.ad.jp/dbget-bin/www_bget?mcc+705479(HMGCR)
MMU: http://www.genome.ad.jp/dbget-bin/www_bget?mmu+15357(Hmgcr)
RNO: http://www.genome.ad.jp/dbget-bin/www_bget?rno+25675(Hmgcr)
CFA: http://www.genome.ad.jp/dbget-bin/www_bget?cfa+479182(HMGCR)
BTA: http://www.genome.ad.jp/dbget-bin/www_bget?bta+407159(HMGCR)
GGA: http://www.genome.ad.jp/dbget-bin/www_bget?gga+395145(RCJMB04_14m24)
SPU: http://www.genome.ad.jp/dbget-bin/www_bget?spu+373355(LOC373355)
DME: http://www.genome.ad.jp/dbget-bin/www_bget?dme+Dmel_CG10367(Hmgcr)
CEL: http://www.genome.ad.jp/dbget-bin/www_bget?cel+F08F8.2
OSA: http://www.genome.ad.jp/dbget-bin/www_bget?osa+4347443
SCE: http://www.genome.ad.jp/dbget-bin/www_bget?sce+YLR450W(HMG2) http://www.genome.ad.jp/dbget-bin/www_bget?sce+YML075C(HMG1)
AGO: http://www.genome.ad.jp/dbget-bin/www_bget?ago+AGOS_AER152W
CGR: http://www.genome.ad.jp/dbget-bin/www_bget?cgr+CAGL0L11506g
SPO: http://www.genome.ad.jp/dbget-bin/www_bget?spo+SPCC162.09c(hmg1)
ANI: http://www.genome.ad.jp/dbget-bin/www_bget?ani+AN3817.2
AFM: http://www.genome.ad.jp/dbget-bin/www_bget?afm+AFUA_1G11230 http://www.genome.ad.jp/dbget-bin/www_bget?afm+AFUA_2G03700
AOR: http://www.genome.ad.jp/dbget-bin/www_bget?aor+AO090103000311 http://www.genome.ad.jp/dbget-bin/www_bget?aor+AO090120000217
CNE: http://www.genome.ad.jp/dbget-bin/www_bget?cne+CNF04830
UMA: http://www.genome.ad.jp/dbget-bin/www_bget?uma+UM03014.1
ECU: http://www.genome.ad.jp/dbget-bin/www_bget?ecu+ECU10_1720
DDI: http://www.genome.ad.jp/dbget-bin/www_bget?ddi+DDB_0191125(hmgA) http://www.genome.ad.jp/dbget-bin/www_bget?ddi+DDB_0215357(hmgB)
TBR: http://www.genome.ad.jp/dbget-bin/www_bget?tbr+Tb927.6.4540
TCR: http://www.genome.ad.jp/dbget-bin/www_bget?tcr+506831.40 http://www.genome.ad.jp/dbget-bin/www_bget?tcr+509167.20
LMA: http://www.genome.ad.jp/dbget-bin/www_bget?lma+LmjF30.3190
VCH: http://www.genome.ad.jp/dbget-bin/www_bget?vch+VCA0723
VCO: http://www.genome.ad.jp/dbget-bin/www_bget?vco+VC0395_0662
VVU: http://www.genome.ad.jp/dbget-bin/www_bget?vvu+VV2_0117
VVY: http://www.genome.ad.jp/dbget-bin/www_bget?vvy+VVA0625
VPA: http://www.genome.ad.jp/dbget-bin/www_bget?vpa+VPA0968
VFI: http://www.genome.ad.jp/dbget-bin/www_bget?vfi+VFA0841
PAT: http://www.genome.ad.jp/dbget-bin/www_bget?pat+Patl_0427
CBU: http://www.genome.ad.jp/dbget-bin/www_bget?cbu+CBU_0030 http://www.genome.ad.jp/dbget-bin/www_bget?cbu+CBU_0610
CBD: http://www.genome.ad.jp/dbget-bin/www_bget?cbd+COXBU7E912_0151 http://www.genome.ad.jp/dbget-bin/www_bget?cbd+COXBU7E912_0622(hmgA)
TCX: http://www.genome.ad.jp/dbget-bin/www_bget?tcx+Tcr_1717
DNO: http://www.genome.ad.jp/dbget-bin/www_bget?dno+DNO_0797
CVI: http://www.genome.ad.jp/dbget-bin/www_bget?cvi+CV_1806
SUS: http://www.genome.ad.jp/dbget-bin/www_bget?sus+Acid_5728 http://www.genome.ad.jp/dbget-bin/www_bget?sus+Acid_6132
SAU: http://www.genome.ad.jp/dbget-bin/www_bget?sau+SA2333(mvaA)
SAV: http://www.genome.ad.jp/dbget-bin/www_bget?sav+SAV2545(mvaA)
SAM: http://www.genome.ad.jp/dbget-bin/www_bget?sam+MW2466(mvaA)
SAB: http://www.genome.ad.jp/dbget-bin/www_bget?sab+SAB2419c(mvaA)
SEP: http://www.genome.ad.jp/dbget-bin/www_bget?sep+SE2109
LWE: http://www.genome.ad.jp/dbget-bin/www_bget?lwe+lwe0819(mvaA)
LLA: http://www.genome.ad.jp/dbget-bin/www_bget?lla+L10433(mvaA)
LLC: http://www.genome.ad.jp/dbget-bin/www_bget?llc+LACR_1664
LLM: http://www.genome.ad.jp/dbget-bin/www_bget?llm+llmg_0931(mvaA)
SPY: http://www.genome.ad.jp/dbget-bin/www_bget?spy+SPy_0880(mvaS.1)
SPM: http://www.genome.ad.jp/dbget-bin/www_bget?spm+spyM18_0941(mvaS1)
SPG: http://www.genome.ad.jp/dbget-bin/www_bget?spg+SpyM3_0599(mvaS.1)
SPS: http://www.genome.ad.jp/dbget-bin/www_bget?sps+SPs1254
SPH: http://www.genome.ad.jp/dbget-bin/www_bget?sph+MGAS10270_Spy0744
SPI: http://www.genome.ad.jp/dbget-bin/www_bget?spi+MGAS10750_Spy0778
SPJ: http://www.genome.ad.jp/dbget-bin/www_bget?spj+MGAS2096_Spy0758
SPK: http://www.genome.ad.jp/dbget-bin/www_bget?spk+MGAS9429_Spy0742
SPA: http://www.genome.ad.jp/dbget-bin/www_bget?spa+M6_Spy0703
SPN: http://www.genome.ad.jp/dbget-bin/www_bget?spn+SP_1726
SAG: http://www.genome.ad.jp/dbget-bin/www_bget?sag+SAG1317
SAN: http://www.genome.ad.jp/dbget-bin/www_bget?san+gbs1387
STC: http://www.genome.ad.jp/dbget-bin/www_bget?stc+str0576(mvaA)
STL: http://www.genome.ad.jp/dbget-bin/www_bget?stl+stu0576(mvaA)
STE: http://www.genome.ad.jp/dbget-bin/www_bget?ste+STER_0620
SSA: http://www.genome.ad.jp/dbget-bin/www_bget?ssa+SSA_0337(mvaA)
LPL: http://www.genome.ad.jp/dbget-bin/www_bget?lpl+lp_0447(mvaA)
LJO: http://www.genome.ad.jp/dbget-bin/www_bget?ljo+LJ1608
LSL: http://www.genome.ad.jp/dbget-bin/www_bget?lsl+LSL_0224
LBR: http://www.genome.ad.jp/dbget-bin/www_bget?lbr+LVIS_0450
LGA: http://www.genome.ad.jp/dbget-bin/www_bget?lga+LGAS_1373
EFA: http://www.genome.ad.jp/dbget-bin/www_bget?efa+EF1364
NFA: http://www.genome.ad.jp/dbget-bin/www_bget?nfa+nfa22110
BGA: http://www.genome.ad.jp/dbget-bin/www_bget?bga+BG0708(mvaA)
SRU: http://www.genome.ad.jp/dbget-bin/www_bget?sru+SRU_2422
FPS: http://www.genome.ad.jp/dbget-bin/www_bget?fps+FP2341
MMP: http://www.genome.ad.jp/dbget-bin/www_bget?mmp+MMP0087(hmgA)
MMQ: http://www.genome.ad.jp/dbget-bin/www_bget?mmq+MmarC5_1589
MAC: http://www.genome.ad.jp/dbget-bin/www_bget?mac+MA3073(hmgA)
MBA: http://www.genome.ad.jp/dbget-bin/www_bget?mba+Mbar_A1972
MMA: http://www.genome.ad.jp/dbget-bin/www_bget?mma+MM_0335
MBU: http://www.genome.ad.jp/dbget-bin/www_bget?mbu+Mbur_1098
MHU: http://www.genome.ad.jp/dbget-bin/www_bget?mhu+Mhun_3004
MEM: http://www.genome.ad.jp/dbget-bin/www_bget?mem+Memar_2365
MBN: http://www.genome.ad.jp/dbget-bin/www_bget?mbn+Mboo_0137
MTH: http://www.genome.ad.jp/dbget-bin/www_bget?mth+MTH562
MST: http://www.genome.ad.jp/dbget-bin/www_bget?mst+Msp_0584(hmgA)
MSI: http://www.genome.ad.jp/dbget-bin/www_bget?msi+Msm_0227
MKA: http://www.genome.ad.jp/dbget-bin/www_bget?mka+MK0355(HMG1)
AFU: http://www.genome.ad.jp/dbget-bin/www_bget?afu+AF1736(mvaA)
HAL: http://www.genome.ad.jp/dbget-bin/www_bget?hal+VNG1875G(mvaA)
HMA: http://www.genome.ad.jp/dbget-bin/www_bget?hma+rrnAC3412(mvaA)
HWA: http://www.genome.ad.jp/dbget-bin/www_bget?hwa+HQ3215A(hmgR)
NPH: http://www.genome.ad.jp/dbget-bin/www_bget?nph+NP0368A(mvaA_2) http://www.genome.ad.jp/dbget-bin/www_bget?nph+NP2422A(mvaA_1)
TAC: http://www.genome.ad.jp/dbget-bin/www_bget?tac+Ta0406m
TVO: http://www.genome.ad.jp/dbget-bin/www_bget?tvo+TVN1168
PTO: http://www.genome.ad.jp/dbget-bin/www_bget?pto+PTO1143
PAB: http://www.genome.ad.jp/dbget-bin/www_bget?pab+PAB2106(mvaA)
PFU: http://www.genome.ad.jp/dbget-bin/www_bget?pfu+PF1848
TKO: http://www.genome.ad.jp/dbget-bin/www_bget?tko+TK0914
RCI: http://www.genome.ad.jp/dbget-bin/www_bget?rci+RCIX1027(hmgA) http://www.genome.ad.jp/dbget-bin/www_bget?rci+RCIX376(hmgA)
APE: http://www.genome.ad.jp/dbget-bin/www_bget?ape+APE_1869
IHO: http://www.genome.ad.jp/dbget-bin/www_bget?iho+Igni_0476
HBU: http://www.genome.ad.jp/dbget-bin/www_bget?hbu+Hbut_1531
SSO: http://www.genome.ad.jp/dbget-bin/www_bget?sso+SSO0531
STO: http://www.genome.ad.jp/dbget-bin/www_bget?sto+ST1352
SAI: http://www.genome.ad.jp/dbget-bin/www_bget?sai+Saci_1359
PAI: http://www.genome.ad.jp/dbget-bin/www_bget?pai+PAE2182
PIS: http://www.genome.ad.jp/dbget-bin/www_bget?pis+Pisl_0814
PCL:
PAS:
Репрезентативные нуклеиновые кислоты и полипептиды мевалонат-киназы
HSA: http://www.genome.ad.jp/dbget-bin/www_bget?hsa+4598(MVK)
MCC: http://www.genome.ad.jp/dbget-bin/www_bget?mcc+707645(MVK)
MMU: http://www.genome.ad.jp/dbget-bin/www_bget?mmu+17855(Mvk)
RNO: http://www.genome.ad.jp/dbget-bin/www_bget?rno+81727(Mvk)
CFA: http://www.genome.ad.jp/dbget-bin/www_bget?cfa+486309(MVK)
BTA: http://www.genome.ad.jp/dbget-bin/www_bget?bta+505792(MVK)
GGA: http://www.genome.ad.jp/dbget-bin/www_bget?gga+768555(MVK)
DRE: http://www.genome.ad.jp/dbget-bin/www_bget?dre+492477(zgc:103473)
SPU: http://www.genome.ad.jp/dbget-bin/www_bget?spu+585785(LOC585785)
DME: http://www.genome.ad.jp/dbget-bin/www_bget?dme+Dmel_CG33671
OSA: http://www.genome.ad.jp/dbget-bin/www_bget?osa+4348331
SCE: http://www.genome.ad.jp/dbget-bin/www_bget?sce+YMR208W(ERG12)
AGO: http://www.genome.ad.jp/dbget-bin/www_bget?ago+AGOS_AER335W
PIC: http://www.genome.ad.jp/dbget-bin/www_bget?pic+PICST_40742(ERG12)
CGR: http://www.genome.ad.jp/dbget-bin/www_bget?cgr+CAGL0F03861g
SPO: http://www.genome.ad.jp/dbget-bin/www_bget?spo+SPAC13G6.11c
MGR: http://www.genome.ad.jp/dbget-bin/www_bget?mgr+MGG_06946
ANI: http://www.genome.ad.jp/dbget-bin/www_bget?ani+AN3869.2
AFM: http://www.genome.ad.jp/dbget-bin/www_bget?afm+AFUA_4G07780
AOR: http://www.genome.ad.jp/dbget-bin/www_bget?aor+AO090023000793
CNE: http://www.genome.ad.jp/dbget-bin/www_bget?cne+CNK01740
ECU: http://www.genome.ad.jp/dbget-bin/www_bget?ecu+ECU09_1780
DDI: http://www.genome.ad.jp/dbget-bin/www_bget?ddi+DDBDRAFT_0168621
TET: http://www.genome.ad.jp/dbget-bin/www_bget?tet+TTHERM_00637680
TBR: http://www.genome.ad.jp/dbget-bin/www_bget?tbr+Tb927.4.4070
TCR: http://www.genome.ad.jp/dbget-bin/www_bget?tcr+436521.9 http://www.genome.ad.jp/dbget-bin/www_bget?tcr+509237.10
LMA: http://www.genome.ad.jp/dbget-bin/www_bget?lma+LmjF31.0560
CBU: http://www.genome.ad.jp/dbget-bin/www_bget?cbu+CBU_0608 http://www.genome.ad.jp/dbget-bin/www_bget?cbu+CBU_0609
CBD: http://www.genome.ad.jp/dbget-bin/www_bget?cbd+COXBU7E912_0620(mvk)
LPN: http://www.genome.ad.jp/dbget-bin/www_bget?lpn+lpg2039
LPF: http://www.genome.ad.jp/dbget-bin/www_bget?lpf+lpl2017
LPP: http://www.genome.ad.jp/dbget-bin/www_bget?lpp+lpp2022
BBA: http://www.genome.ad.jp/dbget-bin/www_bget?bba+Bd1027(lmbP) http://www.genome.ad.jp/dbget-bin/www_bget?bba+Bd1630(mvk)
MXA: http://www.genome.ad.jp/dbget-bin/www_bget?mxa+MXAN_5019(mvk)
OIH: http://www.genome.ad.jp/dbget-bin/www_bget?oih+OB0225
SAU: http://www.genome.ad.jp/dbget-bin/www_bget?sau+SA0547(mvaK1)
SAV: http://www.genome.ad.jp/dbget-bin/www_bget?sav+SAV0590(mvaK1)
SAM: http://www.genome.ad.jp/dbget-bin/www_bget?sam+MW0545(mvaK1)
SAR: http://www.genome.ad.jp/dbget-bin/www_bget?sar+SAR0596(mvaK1)
SAS: http://www.genome.ad.jp/dbget-bin/www_bget?sas+SAS0549
SAC: http://www.genome.ad.jp/dbget-bin/www_bget?sac+SACOL0636(mvk)
SAB: http://www.genome.ad.jp/dbget-bin/www_bget?sab+SAB0540(mvaK1)
SAA: http://www.genome.ad.jp/dbget-bin/www_bget?saa+SAUSA300_0572(mvk)
SAO: http://www.genome.ad.jp/dbget-bin/www_bget?sao+SAOUHSC_00577
SEP: http://www.genome.ad.jp/dbget-bin/www_bget?sep+SE0361
SER: http://www.genome.ad.jp/dbget-bin/www_bget?ser+SERP0238(mvk)
SHA: http://www.genome.ad.jp/dbget-bin/www_bget?sha+SH2402(mvaK1)
SSP: http://www.genome.ad.jp/dbget-bin/www_bget?ssp+SSP2122
LMO: http://www.genome.ad.jp/dbget-bin/www_bget?lmo+lmo0010
LMF: http://www.genome.ad.jp/dbget-bin/www_bget?lmf+LMOf2365_0011
LIN: http://www.genome.ad.jp/dbget-bin/www_bget?lin+lin0010
LWE: http://www.genome.ad.jp/dbget-bin/www_bget?lwe+lwe0011(mvk)
LLA: http://www.genome.ad.jp/dbget-bin/www_bget?lla+L7866(yeaG)
LLC: http://www.genome.ad.jp/dbget-bin/www_bget?llc+LACR_0454
LLM: http://www.genome.ad.jp/dbget-bin/www_bget?llm+llmg_0425(mvk)
SPY: http://www.genome.ad.jp/dbget-bin/www_bget?spy+SPy_0876(mvaK1)
SPZ: http://www.genome.ad.jp/dbget-bin/www_bget?spz+M5005_Spy_0682(mvaK1)
SPM: http://www.genome.ad.jp/dbget-bin/www_bget?spm+spyM18_0937(mvaK1)
SPG: http://www.genome.ad.jp/dbget-bin/www_bget?spg+SpyM3_0595(mvaK1)
SPS: http://www.genome.ad.jp/dbget-bin/www_bget?sps+SPs1258
SPH: http://www.genome.ad.jp/dbget-bin/www_bget?sph+MGAS10270_Spy0740(mvaK1)
SPI: http://www.genome.ad.jp/dbget-bin/www_bget?spi+MGAS10750_Spy0774(mvaK1)
SPJ: http://www.genome.ad.jp/dbget-bin/www_bget?spj+MGAS2096_Spy0753(mvaK1)
SPK: http://www.genome.ad.jp/dbget-bin/www_bget?spk+MGAS9429_Spy0737(mvaK1)
SPF: http://www.genome.ad.jp/dbget-bin/www_bget?spf+SpyM51126(mvaK1)
SPA: http://www.genome.ad.jp/dbget-bin/www_bget?spa+M6_Spy0699
SPB: http://www.genome.ad.jp/dbget-bin/www_bget?spb+M28_Spy0662(mvaK1)
SPN: http://www.genome.ad.jp/dbget-bin/www_bget?spn+SP_0381
SPR: http://www.genome.ad.jp/dbget-bin/www_bget?spr+spr0338(mvk)
SPD: http://www.genome.ad.jp/dbget-bin/www_bget?spd+SPD_0346(mvk)
SAG: http://www.genome.ad.jp/dbget-bin/www_bget?sag+SAG1326
SAN: http://www.genome.ad.jp/dbget-bin/www_bget?san+gbs1396
SAK: http://www.genome.ad.jp/dbget-bin/www_bget?sak+SAK_1357(mvk)
SMU: http://www.genome.ad.jp/dbget-bin/www_bget?smu+SMU.181
STC: http://www.genome.ad.jp/dbget-bin/www_bget?stc+str0559(mvaK1)
STL: http://www.genome.ad.jp/dbget-bin/www_bget?stl+stu0559(mvaK1)
STE: http://www.genome.ad.jp/dbget-bin/www_bget?ste+STER_0598
SSA: http://www.genome.ad.jp/dbget-bin/www_bget?ssa+SSA_0333(mvaK1)
SSU: http://www.genome.ad.jp/dbget-bin/www_bget?ssu+SSU05_0289
SSV: http://www.genome.ad.jp/dbget-bin/www_bget?ssv+SSU98_0285
SGO: http://www.genome.ad.jp/dbget-bin/www_bget?sgo+SGO_0239(mvk)
LPL: http://www.genome.ad.jp/dbget-bin/www_bget?lpl+lp_1735(mvaK1)
LJO: http://www.genome.ad.jp/dbget-bin/www_bget?ljo+LJ1205
LAC: http://www.genome.ad.jp/dbget-bin/www_bget?lac+LBA1167(mvaK)
LSA: http://www.genome.ad.jp/dbget-bin/www_bget?lsa+LSA0908(mvaK1)
LSL: http://www.genome.ad.jp/dbget-bin/www_bget?lsl+LSL_0685(eRG)
LDB: http://www.genome.ad.jp/dbget-bin/www_bget?ldb+Ldb0999(mvk)
LBU: http://www.genome.ad.jp/dbget-bin/www_bget?lbu+LBUL_0906
LBR: http://www.genome.ad.jp/dbget-bin/www_bget?lbr+LVIS_0858
LCA: http://www.genome.ad.jp/dbget-bin/www_bget?lca+LSEI_1491
LGA: http://www.genome.ad.jp/dbget-bin/www_bget?lga+LGAS_1033
LRE: http://www.genome.ad.jp/dbget-bin/www_bget?lre+Lreu_0915
PPE: http://www.genome.ad.jp/dbget-bin/www_bget?ppe+PEPE_0927
EFA: http://www.genome.ad.jp/dbget-bin/www_bget?efa+EF0904(mvk)
OOE: http://www.genome.ad.jp/dbget-bin/www_bget?ooe+OEOE_1100
LME: http://www.genome.ad.jp/dbget-bin/www_bget?lme+LEUM_1385
NFA: http://www.genome.ad.jp/dbget-bin/www_bget?nfa+nfa22070
BGA: http://www.genome.ad.jp/dbget-bin/www_bget?bga+BG0711
BAF: http://www.genome.ad.jp/dbget-bin/www_bget?baf+BAPKO_0732
FPS: http://www.genome.ad.jp/dbget-bin/www_bget?fps+FP0313
MMP: http://www.genome.ad.jp/dbget-bin/www_bget?mmp+MMP1335
MAE: http://www.genome.ad.jp/dbget-bin/www_bget?mae+Maeo_0775
MAC: http://www.genome.ad.jp/dbget-bin/www_bget?mac+MA0602(mvk)
MBA: http://www.genome.ad.jp/dbget-bin/www_bget?mba+Mbar_A1421
MMA: http://www.genome.ad.jp/dbget-bin/www_bget?mma+MM_1762
MBU: http://www.genome.ad.jp/dbget-bin/www_bget?mbu+Mbur_2395
MHU: http://www.genome.ad.jp/dbget-bin/www_bget?mhu+Mhun_2890
MEM: http://www.genome.ad.jp/dbget-bin/www_bget?mem+Memar_1812
MBN: http://www.genome.ad.jp/dbget-bin/www_bget?mbn+Mboo_2213
MST: http://www.genome.ad.jp/dbget-bin/www_bget?mst+Msp_0858(mvk)
MSI: http://www.genome.ad.jp/dbget-bin/www_bget?msi+Msm_1439
MKA: http://www.genome.ad.jp/dbget-bin/www_bget?mka+MK0993(ERG12)
HAL: http://www.genome.ad.jp/dbget-bin/www_bget?hal+VNG1145G(mvk)
HMA: http://www.genome.ad.jp/dbget-bin/www_bget?hma+rrnAC0077(mvk)
HWA: http://www.genome.ad.jp/dbget-bin/www_bget?hwa+HQ2925A(mvk)
NPH: http://www.genome.ad.jp/dbget-bin/www_bget?nph+NP2850A(mvk)
PTO: http://www.genome.ad.jp/dbget-bin/www_bget?pto+PTO1352
PHO: http://www.genome.ad.jp/dbget-bin/www_bget?pho+PH1625
PAB: http://www.genome.ad.jp/dbget-bin/www_bget?pab+PAB0372(mvk)
PFU: http://www.genome.ad.jp/dbget-bin/www_bget?pfu+PF1637(mvk)
TKO: http://www.genome.ad.jp/dbget-bin/www_bget?tko+TK1474
RCI: http://www.genome.ad.jp/dbget-bin/www_bget?rci+LRC399(mvk)
APE: http://www.genome.ad.jp/dbget-bin/www_bget?ape+APE_2439
HBU: http://www.genome.ad.jp/dbget-bin/www_bget?hbu+Hbut_0877
SSO: http://www.genome.ad.jp/dbget-bin/www_bget?sso+SSO0383
STO: http://www.genome.ad.jp/dbget-bin/www_bget?sto+ST2185
SAI: http://www.genome.ad.jp/dbget-bin/www_bget?sai+Saci_2365(mvk)
MSE: http://www.genome.ad.jp/dbget-bin/www_bget?mse+Msed_1602
PAI: http://www.genome.ad.jp/dbget-bin/www_bget?pai+PAE3108
PIS: http://www.genome.ad.jp/dbget-bin/www_bget?pis+Pisl_0467
PCL:
Репрезентативные нуклеиновые кислоты и полипептиды фосфомевалонат-киназы
HSA: http://www.genome.ad.jp/dbget-bin/www_bget?hsa+10654(PMVK)
PTR: http://www.genome.ad.jp/dbget-bin/www_bget?ptr+457350(PMVK)
MCC: http://www.genome.ad.jp/dbget-bin/www_bget?mcc+717014(PMVK)
MMU: http://www.genome.ad.jp/dbget-bin/www_bget?mmu+68603(Pmvk)
CFA: http://www.genome.ad.jp/dbget-bin/www_bget?cfa+612251(PMVK)
BTA: http://www.genome.ad.jp/dbget-bin/www_bget?bta+513533(PMVK)
DME: http://www.genome.ad.jp/dbget-bin/www_bget?dme+Dmel_CG10268
ATH: http://www.genome.ad.jp/dbget-bin/www_bget?ath+AT1G31910
OSA: http://www.genome.ad.jp/dbget-bin/www_bget?osa+4332275
SCE: http://www.genome.ad.jp/dbget-bin/www_bget?sce+YMR220W(ERG8)
AGO: http://www.genome.ad.jp/dbget-bin/www_bget?ago+AGOS_AER354W
PIC: http://www.genome.ad.jp/dbget-bin/www_bget?pic+PICST_52257(ERG8)
CGR: http://www.genome.ad.jp/dbget-bin/www_bget?cgr+CAGL0F03993g
SPO: http://www.genome.ad.jp/dbget-bin/www_bget?spo+SPAC343.01c
MGR: http://www.genome.ad.jp/dbget-bin/www_bget?mgr+MGG_05812
ANI: http://www.genome.ad.jp/dbget-bin/www_bget?ani+AN2311.2
AFM: http://www.genome.ad.jp/dbget-bin/www_bget?afm+AFUA_5G10680
AOR: http://www.genome.ad.jp/dbget-bin/www_bget?aor+AO090010000471
CNE: http://www.genome.ad.jp/dbget-bin/www_bget?cne+CNM00100
UMA: http://www.genome.ad.jp/dbget-bin/www_bget?uma+UM00760.1
DDI: http://www.genome.ad.jp/dbget-bin/www_bget?ddi+DDBDRAFT_0184512
TBR: http://www.genome.ad.jp/dbget-bin/www_bget?tbr+Tb09.160.3690
TCR: http://www.genome.ad.jp/dbget-bin/www_bget?tcr+507913.20 http://www.genome.ad.jp/dbget-bin/www_bget?tcr+508277.140
LMA: http://www.genome.ad.jp/dbget-bin/www_bget?lma+LmjF15.1460
MXA: http://www.genome.ad.jp/dbget-bin/www_bget?mxa+MXAN_5017
OIH: http://www.genome.ad.jp/dbget-bin/www_bget?oih+OB0227
SAU: http://www.genome.ad.jp/dbget-bin/www_bget?sau+SA0549(mvaK2)
SAV: http://www.genome.ad.jp/dbget-bin/www_bget?sav+SAV0592(mvaK2)
SAM: http://www.genome.ad.jp/dbget-bin/www_bget?sam+MW0547(mvaK2)
SAR: http://www.genome.ad.jp/dbget-bin/www_bget?sar+SAR0598(mvaK2)
SAS: http://www.genome.ad.jp/dbget-bin/www_bget?sas+SAS0551
SAC: http://www.genome.ad.jp/dbget-bin/www_bget?sac+SACOL0638
SAB: http://www.genome.ad.jp/dbget-bin/www_bget?sab+SAB0542(mvaK2)
SAA: http://www.genome.ad.jp/dbget-bin/www_bget?saa+SAUSA300_0574
SAO: http://www.genome.ad.jp/dbget-bin/www_bget?sao+SAOUHSC_00579
SAJ: http://www.genome.ad.jp/dbget-bin/www_bget?saj+SaurJH9_0615
SEP: http://www.genome.ad.jp/dbget-bin/www_bget?sep+SE0363
SER: http://www.genome.ad.jp/dbget-bin/www_bget?ser+SERP0240
SHA: http://www.genome.ad.jp/dbget-bin/www_bget?sha+SH2400(mvaK2)
SSP: http://www.genome.ad.jp/dbget-bin/www_bget?ssp+SSP2120
LMO: http://www.genome.ad.jp/dbget-bin/www_bget?lmo+lmo0012
LMF: http://www.genome.ad.jp/dbget-bin/www_bget?lmf+LMOf2365_0013
LIN: http://www.genome.ad.jp/dbget-bin/www_bget?lin+lin0012
LWE: http://www.genome.ad.jp/dbget-bin/www_bget?lwe+lwe0013
LLA: http://www.genome.ad.jp/dbget-bin/www_bget?lla+L10014(yebA)
LLC: http://www.genome.ad.jp/dbget-bin/www_bget?llc+LACR_0456
LLM: http://www.genome.ad.jp/dbget-bin/www_bget?llm+llmg_0427
SPY: http://www.genome.ad.jp/dbget-bin/www_bget?spy+SPy_0878(mvaK2)
SPZ: http://www.genome.ad.jp/dbget-bin/www_bget?spz+M5005_Spy_0684(mvaK2)
SPM: http://www.genome.ad.jp/dbget-bin/www_bget?spm+spyM18_0939
SPG: http://www.genome.ad.jp/dbget-bin/www_bget?spg+SpyM3_0597(mvaK2)
SPS: http://www.genome.ad.jp/dbget-bin/www_bget?sps+SPs1256
SPH: http://www.genome.ad.jp/dbget-bin/www_bget?sph+MGAS10270_Spy0742(mvaK2)
SPI: http://www.genome.ad.jp/dbget-bin/www_bget?spi+MGAS10750_Spy0776(mvaK2)
SPJ: http://www.genome.ad.jp/dbget-bin/www_bget?spj+MGAS2096_Spy0755(mvaK2)
SPK: http://www.genome.ad.jp/dbget-bin/www_bget?spk+MGAS9429_Spy0739(mvaK2)
SPF: http://www.genome.ad.jp/dbget-bin/www_bget?spf+SpyM51124(mvaK2)
SPA: http://www.genome.ad.jp/dbget-bin/www_bget?spa+M6_Spy0701
SPB: http://www.genome.ad.jp/dbget-bin/www_bget?spb+M28_Spy0664(mvaK2)
SPN: http://www.genome.ad.jp/dbget-bin/www_bget?spn+SP_0383
SPR: http://www.genome.ad.jp/dbget-bin/www_bget?spr+spr0340(mvaK2)
SPD: http://www.genome.ad.jp/dbget-bin/www_bget?spd+SPD_0348(mvaK2)
SAG: http://www.genome.ad.jp/dbget-bin/www_bget?sag+SAG1324
SAN: http://www.genome.ad.jp/dbget-bin/www_bget?san+gbs1394
SAK: http://www.genome.ad.jp/dbget-bin/www_bget?sak+SAK_1355
SMU: http://www.genome.ad.jp/dbget-bin/www_bget?smu+SMU.938
STC: http://www.genome.ad.jp/dbget-bin/www_bget?stc+str0561(mvaK2)
STL: http://www.genome.ad.jp/dbget-bin/www_bget?stl+stu0561(mvaK2)
STE: http://www.genome.ad.jp/dbget-bin/www_bget?ste+STER_0600
SSA: http://www.genome.ad.jp/dbget-bin/www_bget?ssa+SSA_0335(mvaK2)
SSU: http://www.genome.ad.jp/dbget-bin/www_bget?ssu+SSU05_0291
SSV: http://www.genome.ad.jp/dbget-bin/www_bget?ssv+SSU98_0287
SGO: http://www.genome.ad.jp/dbget-bin/www_bget?sgo+SGO_0241
LPL: http://www.genome.ad.jp/dbget-bin/www_bget?lpl+lp_1733(mvaK2)
LJO: http://www.genome.ad.jp/dbget-bin/www_bget?ljo+LJ1207
LAC: http://www.genome.ad.jp/dbget-bin/www_bget?lac+LBA1169
LSA: http://www.genome.ad.jp/dbget-bin/www_bget?lsa+LSA0906(mvaK2)
LSL: http://www.genome.ad.jp/dbget-bin/www_bget?lsl+LSL_0683
LDB: http://www.genome.ad.jp/dbget-bin/www_bget?ldb+Ldb0997(mvaK)
LBU: http://www.genome.ad.jp/dbget-bin/www_bget?lbu+LBUL_0904
LBR: http://www.genome.ad.jp/dbget-bin/www_bget?lbr+LVIS_0860
LCA: http://www.genome.ad.jp/dbget-bin/www_bget?lca+LSEI_1092
LGA: http://www.genome.ad.jp/dbget-bin/www_bget?lga+LGAS_1035
LRE: http://www.genome.ad.jp/dbget-bin/www_bget?lre+Lreu_0913
PPE: http://www.genome.ad.jp/dbget-bin/www_bget?ppe+PEPE_0925
EFA: http://www.genome.ad.jp/dbget-bin/www_bget?efa+EF0902
NFA: http://www.genome.ad.jp/dbget-bin/www_bget?nfa+nfa22090
BGA: http://www.genome.ad.jp/dbget-bin/www_bget?bga+BG0710
BAF: http://www.genome.ad.jp/dbget-bin/www_bget?baf+BAPKO_0731
NPH: http://www.genome.ad.jp/dbget-bin/www_bget?nph+NP2852A
SSO: http://www.genome.ad.jp/dbget-bin/www_bget?sso+SSO2988
STO:
SAI:
Репрезентативные нуклеиновые кислоты и полипептиды дифосфомевалонат-декарбоксилазы
HSA: http://www.genome.ad.jp/dbget-bin/www_bget?hsa+4597(MVD)
PTR: http://www.genome.ad.jp/dbget-bin/www_bget?ptr+468069(MVD)
MCC: http://www.genome.ad.jp/dbget-bin/www_bget?mcc+696865(MVD)
MMU: http://www.genome.ad.jp/dbget-bin/www_bget?mmu+192156(Mvd)
RNO: http://www.genome.ad.jp/dbget-bin/www_bget?rno+81726(Mvd)
CFA: http://www.genome.ad.jp/dbget-bin/www_bget?cfa+489663(MVD)
GGA: http://www.genome.ad.jp/dbget-bin/www_bget?gga+425359(MVD)
DME: http://www.genome.ad.jp/dbget-bin/www_bget?dme+Dmel_CG8239
SCE: http://www.genome.ad.jp/dbget-bin/www_bget?sce+YNR043W(MVD1)
AGO: http://www.genome.ad.jp/dbget-bin/www_bget?ago+AGOS_AGL232C
PIC: http://www.genome.ad.jp/dbget-bin/www_bget?pic+PICST_90752
CGR: http://www.genome.ad.jp/dbget-bin/www_bget?cgr+CAGL0C03630g
SPO: http://www.genome.ad.jp/dbget-bin/www_bget?spo+SPAC24C9.03
MGR: http://www.genome.ad.jp/dbget-bin/www_bget?mgr+MGG_09750
ANI: http://www.genome.ad.jp/dbget-bin/www_bget?ani+AN4414.2
AFM: http://www.genome.ad.jp/dbget-bin/www_bget?afm+AFUA_4G07130
AOR: http://www.genome.ad.jp/dbget-bin/www_bget?aor+AO090023000862
CNE: http://www.genome.ad.jp/dbget-bin/www_bget?cne+CNL04950
UMA: http://www.genome.ad.jp/dbget-bin/www_bget?uma+UM05179.1
DDI: http://www.genome.ad.jp/dbget-bin/www_bget?ddi+DDBDRAFT_0218058
TET: http://www.genome.ad.jp/dbget-bin/www_bget?tet+TTHERM_00849200
TBR: http://www.genome.ad.jp/dbget-bin/www_bget?tbr+Tb10.05.0010 http://www.genome.ad.jp/dbget-bin/www_bget?tbr+Tb10.61.2745
TCR: http://www.genome.ad.jp/dbget-bin/www_bget?tcr+507993.330 http://www.genome.ad.jp/dbget-bin/www_bget?tcr+511281.40
LMA: http://www.genome.ad.jp/dbget-bin/www_bget?lma+LmjF18.0020
CBU: http://www.genome.ad.jp/dbget-bin/www_bget?cbu+CBU_0607(mvaD)
CBD: http://www.genome.ad.jp/dbget-bin/www_bget?cbd+COXBU7E912_0619(mvaD)
LPN: http://www.genome.ad.jp/dbget-bin/www_bget?lpn+lpg2040
LPF: http://www.genome.ad.jp/dbget-bin/www_bget?lpf+lpl2018
LPP: http://www.genome.ad.jp/dbget-bin/www_bget?lpp+lpp2023
TCX: http://www.genome.ad.jp/dbget-bin/www_bget?tcx+Tcr_1734
DNO: http://www.genome.ad.jp/dbget-bin/www_bget?dno+DNO_0504(mvaD)
BBA: http://www.genome.ad.jp/dbget-bin/www_bget?bba+Bd1629
MXA: http://www.genome.ad.jp/dbget-bin/www_bget?mxa+MXAN_5018(mvaD)
OIH: http://www.genome.ad.jp/dbget-bin/www_bget?oih+OB0226
SAU: http://www.genome.ad.jp/dbget-bin/www_bget?sau+SA0548(mvaD)
SAV: http://www.genome.ad.jp/dbget-bin/www_bget?sav+SAV0591(mvaD)
SAM: http://www.genome.ad.jp/dbget-bin/www_bget?sam+MW0546(mvaD)
SAR: http://www.genome.ad.jp/dbget-bin/www_bget?sar+SAR0597(mvaD)
SAS: http://www.genome.ad.jp/dbget-bin/www_bget?sas+SAS0550
SAC: http://www.genome.ad.jp/dbget-bin/www_bget?sac+SACOL0637(mvaD)
SAB: http://www.genome.ad.jp/dbget-bin/www_bget?sab+SAB0541(mvaD)
SAA: http://www.genome.ad.jp/dbget-bin/www_bget?saa+SAUSA300_0573(mvaD)
SAO: http://www.genome.ad.jp/dbget-bin/www_bget?sao+SAOUHSC_00578
SAJ: http://www.genome.ad.jp/dbget-bin/www_bget?saj+SaurJH9_0614
SAH: http://www.genome.ad.jp/dbget-bin/www_bget?sah+SaurJH1_0629
SEP: http://www.genome.ad.jp/dbget-bin/www_bget?sep+SE0362
SER: http://www.genome.ad.jp/dbget-bin/www_bget?ser+SERP0239(mvaD)
SHA: http://www.genome.ad.jp/dbget-bin/www_bget?sha+SH2401(mvaD)
SSP: http://www.genome.ad.jp/dbget-bin/www_bget?ssp+SSP2121
LMO: http://www.genome.ad.jp/dbget-bin/www_bget?lmo+lmo0011
LMF: http://www.genome.ad.jp/dbget-bin/www_bget?lmf+LMOf2365_0012(mvaD)
LIN: http://www.genome.ad.jp/dbget-bin/www_bget?lin+lin0011
LWE: http://www.genome.ad.jp/dbget-bin/www_bget?lwe+lwe0012(mvaD)
LLA: http://www.genome.ad.jp/dbget-bin/www_bget?lla+L9089(yeaH)
LLC: http://www.genome.ad.jp/dbget-bin/www_bget?llc+LACR_0455
LLM: http://www.genome.ad.jp/dbget-bin/www_bget?llm+llmg_0426(mvaD)
SPY: http://www.genome.ad.jp/dbget-bin/www_bget?spy+SPy_0877(mvaD)
SPZ: http://www.genome.ad.jp/dbget-bin/www_bget?spz+M5005_Spy_0683(mvaD)
SPM: http://www.genome.ad.jp/dbget-bin/www_bget?spm+spyM18_0938(mvd)
SPG: http://www.genome.ad.jp/dbget-bin/www_bget?spg+SpyM3_0596(mvaD)
SPS: http://www.genome.ad.jp/dbget-bin/www_bget?sps+SPs1257
SPH: http://www.genome.ad.jp/dbget-bin/www_bget?sph+MGAS10270_Spy0741(mvaD)
SPI: http://www.genome.ad.jp/dbget-bin/www_bget?spi+MGAS10750_Spy0775(mvaD)
SPJ: http://www.genome.ad.jp/dbget-bin/www_bget?spj+MGAS2096_Spy0754(mvaD)
SPK: http://www.genome.ad.jp/dbget-bin/www_bget?spk+MGAS9429_Spy0738(mvaD)
SPF: http://www.genome.ad.jp/dbget-bin/www_bget?spf+SpyM51125(mvaD)
SPA: http://www.genome.ad.jp/dbget-bin/www_bget?spa+M6_Spy0700
SPB: http://www.genome.ad.jp/dbget-bin/www_bget?spb+M28_Spy0663(mvaD)
SPN: http://www.genome.ad.jp/dbget-bin/www_bget?spn+SP_0382
SPR: http://www.genome.ad.jp/dbget-bin/www_bget?spr+spr0339(mvd1)
SPD: http://www.genome.ad.jp/dbget-bin/www_bget?spd+SPD_0347(mvaD)
SAG: http://www.genome.ad.jp/dbget-bin/www_bget?sag+SAG1325(mvaD)
SAN: http://www.genome.ad.jp/dbget-bin/www_bget?san+gbs1395
SAK: http://www.genome.ad.jp/dbget-bin/www_bget?sak+SAK_1356(mvaD)
SMU: http://www.genome.ad.jp/dbget-bin/www_bget?smu+SMU.937
STC: http://www.genome.ad.jp/dbget-bin/www_bget?stc+str0560(mvaD)
STL: http://www.genome.ad.jp/dbget-bin/www_bget?stl+stu0560(mvaD)
STE: http://www.genome.ad.jp/dbget-bin/www_bget?ste+STER_0599
SSA: http://www.genome.ad.jp/dbget-bin/www_bget?ssa+SSA_0334(mvaD)
SSU: http://www.genome.ad.jp/dbget-bin/www_bget?ssu+SSU05_0290
SSV: http://www.genome.ad.jp/dbget-bin/www_bget?ssv+SSU98_0286
SGO: http://www.genome.ad.jp/dbget-bin/www_bget?sgo+SGO_0240(mvaD)
LPL: http://www.genome.ad.jp/dbget-bin/www_bget?lpl+lp_1734(mvaD)
LJO: http://www.genome.ad.jp/dbget-bin/www_bget?ljo+LJ1206
LAC: http://www.genome.ad.jp/dbget-bin/www_bget?lac+LBA1168(mvaD)
LSA: http://www.genome.ad.jp/dbget-bin/www_bget?lsa+LSA0907(mvaD)
LSL: http://www.genome.ad.jp/dbget-bin/www_bget?lsl+LSL_0684
LDB: http://www.genome.ad.jp/dbget-bin/www_bget?ldb+Ldb0998(mvaD)
LBU: http://www.genome.ad.jp/dbget-bin/www_bget?lbu+LBUL_0905
LBR: http://www.genome.ad.jp/dbget-bin/www_bget?lbr+LVIS_0859
LCA: http://www.genome.ad.jp/dbget-bin/www_bget?lca+LSEI_1492
LGA: http://www.genome.ad.jp/dbget-bin/www_bget?lga+LGAS_1034
LRE: http://www.genome.ad.jp/dbget-bin/www_bget?lre+Lreu_0914
PPE: http://www.genome.ad.jp/dbget-bin/www_bget?ppe+PEPE_0926
EFA: http://www.genome.ad.jp/dbget-bin/www_bget?efa+EF0903(mvaD)
LME: http://www.genome.ad.jp/dbget-bin/www_bget?lme+LEUM_1386
NFA: http://www.genome.ad.jp/dbget-bin/www_bget?nfa+nfa22080
BBU: http://www.genome.ad.jp/dbget-bin/www_bget?bbu+BB0686
BGA: http://www.genome.ad.jp/dbget-bin/www_bget?bga+BG0709
BAF: http://www.genome.ad.jp/dbget-bin/www_bget?baf+BAPKO_0730
GFO: http://www.genome.ad.jp/dbget-bin/www_bget?gfo+GFO_3632
FPS: http://www.genome.ad.jp/dbget-bin/www_bget?fps+FP0310(mvaD)
HAU: http://www.genome.ad.jp/dbget-bin/www_bget?hau+Haur_1612
HAL: http://www.genome.ad.jp/dbget-bin/www_bget?hal+VNG0593G(dmd)
HMA: http://www.genome.ad.jp/dbget-bin/www_bget?hma+rrnAC1489(dmd)
HWA: http://www.genome.ad.jp/dbget-bin/www_bget?hwa+HQ1525A(mvaD)
NPH: http://www.genome.ad.jp/dbget-bin/www_bget?nph+NP1580A(mvaD)
PTO: http://www.genome.ad.jp/dbget-bin/www_bget?pto+PTO0478 http://www.genome.ad.jp/dbget-bin/www_bget?pto+PTO1356
SSO: http://www.genome.ad.jp/dbget-bin/www_bget?sso+SSO2989
STO: http://www.genome.ad.jp/dbget-bin/www_bget?sto+ST0977
SAI: http://www.genome.ad.jp/dbget-bin/www_bget?sai+Saci_1245(mvd)
MSE:
Репрезентативные нуклеиновые кислоты и полипептиды изопентенил-дифосфат-дельта-изомеразы (IDI)
HSA: http://www.genome.ad.jp/dbget-bin/www_bget?hsa+3422(IDI1) http://www.genome.ad.jp/dbget-bin/www_bget?hsa+91734(IDI2)
PTR: http://www.genome.ad.jp/dbget-bin/www_bget?ptr+450262(IDI2) http://www.genome.ad.jp/dbget-bin/www_bget?ptr+450263(IDI1)
MCC: http://www.genome.ad.jp/dbget-bin/www_bget?mcc+710052(LOC710052) http://www.genome.ad.jp/dbget-bin/www_bget?mcc+721730(LOC721730)
MMU: http://www.genome.ad.jp/dbget-bin/www_bget?mmu+319554(Idi1)
RNO: http://www.genome.ad.jp/dbget-bin/www_bget?rno+89784(Idi1)
GGA: http://www.genome.ad.jp/dbget-bin/www_bget?gga+420459(IDI1)
XLA: http://www.genome.ad.jp/dbget-bin/www_bget?xla+494671(LOC494671)
XTR: http://www.genome.ad.jp/dbget-bin/www_bget?xtr+496783(idi2)
SPU: http://www.genome.ad.jp/dbget-bin/www_bget?spu+586184(LOC586184)
CEL: http://www.genome.ad.jp/dbget-bin/www_bget?cel+K06H7.9(idi-1)
ATH: http://www.genome.ad.jp/dbget-bin/www_bget?ath+AT3G02780(IPP2)
OSA: http://www.genome.ad.jp/dbget-bin/www_bget?osa+4338791 http://www.genome.ad.jp/dbget-bin/www_bget?osa+4343523
CME: http://www.genome.ad.jp/dbget-bin/www_bget?cme+CMB062C
SCE: http://www.genome.ad.jp/dbget-bin/www_bget?sce+YPL117C(IDI1)
AGO: http://www.genome.ad.jp/dbget-bin/www_bget?ago+AGOS_ADL268C
PIC: http://www.genome.ad.jp/dbget-bin/www_bget?pic+PICST_68990(IDI1)
CGR: http://www.genome.ad.jp/dbget-bin/www_bget?cgr+CAGL0J06952g
SPO: http://www.genome.ad.jp/dbget-bin/www_bget?spo+SPBC106.15(idi1)
ANI: http://www.genome.ad.jp/dbget-bin/www_bget?ani+AN0579.2
AFM: http://www.genome.ad.jp/dbget-bin/www_bget?afm+AFUA_6G11160
AOR: http://www.genome.ad.jp/dbget-bin/www_bget?aor+AO090023000500
CNE: http://www.genome.ad.jp/dbget-bin/www_bget?cne+CNA02550
UMA: http://www.genome.ad.jp/dbget-bin/www_bget?uma+UM04838.1
ECU: http://www.genome.ad.jp/dbget-bin/www_bget?ecu+ECU02_0230
DDI: http://www.genome.ad.jp/dbget-bin/www_bget?ddi+DDB_0191342(ipi)
TET: http://www.genome.ad.jp/dbget-bin/www_bget?tet+TTHERM_00237280 http://www.genome.ad.jp/dbget-bin/www_bget?tet+TTHERM_00438860
TBR: http://www.genome.ad.jp/dbget-bin/www_bget?tbr+Tb09.211.0700
TCR: http://www.genome.ad.jp/dbget-bin/www_bget?tcr+408799.19 http://www.genome.ad.jp/dbget-bin/www_bget?tcr+510431.10
LMA: http://www.genome.ad.jp/dbget-bin/www_bget?lma+LmjF35.5330
EHI: http://www.genome.ad.jp/dbget-bin/www_bget?ehi+46.t00025
ECO: http://www.genome.ad.jp/dbget-bin/www_bget?eco+b2889(idi)
ECJ: http://www.genome.ad.jp/dbget-bin/www_bget?ecj+JW2857(idi)
ECE: http://www.genome.ad.jp/dbget-bin/www_bget?ece+Z4227
ECS: http://www.genome.ad.jp/dbget-bin/www_bget?ecs+ECs3761
ECC: http://www.genome.ad.jp/dbget-bin/www_bget?ecc+c3467
ECI: http://www.genome.ad.jp/dbget-bin/www_bget?eci+UTI89_C3274
ECP: http://www.genome.ad.jp/dbget-bin/www_bget?ecp+ECP_2882
ECV: http://www.genome.ad.jp/dbget-bin/www_bget?ecv+APECO1_3638
ECW: http://www.genome.ad.jp/dbget-bin/www_bget?ecw+EcE24377A_3215(idi)
ECX: http://www.genome.ad.jp/dbget-bin/www_bget?ecx+EcHS_A3048
STY: http://www.genome.ad.jp/dbget-bin/www_bget?sty+STY3195
STT: http://www.genome.ad.jp/dbget-bin/www_bget?stt+t2957
SPT: http://www.genome.ad.jp/dbget-bin/www_bget?spt+SPA2907(idi)
SEC: http://www.genome.ad.jp/dbget-bin/www_bget?sec+SC2979(idi)
STM: http://www.genome.ad.jp/dbget-bin/www_bget?stm+STM3039(idi)
SFL: http://www.genome.ad.jp/dbget-bin/www_bget?sfl+SF2875(idi)
SFX: http://www.genome.ad.jp/dbget-bin/www_bget?sfx+S3074
SFV: http://www.genome.ad.jp/dbget-bin/www_bget?sfv+SFV_2937
SSN: http://www.genome.ad.jp/dbget-bin/www_bget?ssn+SSON_3042 http://www.genome.ad.jp/dbget-bin/www_bget?ssn+SSON_3489(yhfK)
SBO: http://www.genome.ad.jp/dbget-bin/www_bget?sbo+SBO_3103
SDY: http://www.genome.ad.jp/dbget-bin/www_bget?sdy+SDY_3193
ECA: http://www.genome.ad.jp/dbget-bin/www_bget?eca+ECA2789
PLU: http://www.genome.ad.jp/dbget-bin/www_bget?plu+plu3987
ENT: http://www.genome.ad.jp/dbget-bin/www_bget?ent+Ent638_3307
SPE: http://www.genome.ad.jp/dbget-bin/www_bget?spe+Spro_2201
VPA: http://www.genome.ad.jp/dbget-bin/www_bget?vpa+VPA0278
VFI: http://www.genome.ad.jp/dbget-bin/www_bget?vfi+VF0403
PPR: http://www.genome.ad.jp/dbget-bin/www_bget?ppr+PBPRA0469(mvaD)
PEN: http://www.genome.ad.jp/dbget-bin/www_bget?pen+PSEEN4850
CBU: http://www.genome.ad.jp/dbget-bin/www_bget?cbu+CBU_0607(mvaD)
CBD: http://www.genome.ad.jp/dbget-bin/www_bget?cbd+COXBU7E912_0619(mvaD)
LPN: http://www.genome.ad.jp/dbget-bin/www_bget?lpn+lpg2051
LPF: http://www.genome.ad.jp/dbget-bin/www_bget?lpf+lpl2029
LPP: http://www.genome.ad.jp/dbget-bin/www_bget?lpp+lpp2034
TCX: http://www.genome.ad.jp/dbget-bin/www_bget?tcx+Tcr_1718
HHA: http://www.genome.ad.jp/dbget-bin/www_bget?hha+Hhal_1623
DNO: http://www.genome.ad.jp/dbget-bin/www_bget?dno+DNO_0798
EBA: http://www.genome.ad.jp/dbget-bin/www_bget?eba+ebA5678 http://www.genome.ad.jp/dbget-bin/www_bget?eba+p2A143
DVU: http://www.genome.ad.jp/dbget-bin/www_bget?dvu+DVU1679(idi)
DDE: http://www.genome.ad.jp/dbget-bin/www_bget?dde+Dde_1991
LIP: http://www.genome.ad.jp/dbget-bin/www_bget?lip+LI1134
BBA: http://www.genome.ad.jp/dbget-bin/www_bget?bba+Bd1626
AFW: http://www.genome.ad.jp/dbget-bin/www_bget?afw+Anae109_4082
MXA: http://www.genome.ad.jp/dbget-bin/www_bget?mxa+MXAN_5021(fni)
RPR: http://www.genome.ad.jp/dbget-bin/www_bget?rpr+RP452
RTY: http://www.genome.ad.jp/dbget-bin/www_bget?rty+RT0439(idi)
RCO: http://www.genome.ad.jp/dbget-bin/www_bget?rco+RC0744
RFE: http://www.genome.ad.jp/dbget-bin/www_bget?rfe+RF_0785(fni)
RBE: http://www.genome.ad.jp/dbget-bin/www_bget?rbe+RBE_0731(fni)
RAK: http://www.genome.ad.jp/dbget-bin/www_bget?rak+A1C_04190
RBO: http://www.genome.ad.jp/dbget-bin/www_bget?rbo+A1I_04755
RCM: http://www.genome.ad.jp/dbget-bin/www_bget?rcm+A1E_02555
RRI: http://www.genome.ad.jp/dbget-bin/www_bget?rri+A1G_04195
MLO: http://www.genome.ad.jp/dbget-bin/www_bget?mlo+mlr6371
RET: http://www.genome.ad.jp/dbget-bin/www_bget?ret+RHE_PD00245(ypd00046)
XAU: http://www.genome.ad.jp/dbget-bin/www_bget?xau+Xaut_4134
SIL: http://www.genome.ad.jp/dbget-bin/www_bget?sil+SPO0131
SIT: http://www.genome.ad.jp/dbget-bin/www_bget?sit+TM1040_3442
RSP: http://www.genome.ad.jp/dbget-bin/www_bget?rsp+RSP_0276
RSH: http://www.genome.ad.jp/dbget-bin/www_bget?rsh+Rsph17029_1919
RSQ: http://www.genome.ad.jp/dbget-bin/www_bget?rsq+Rsph17025_1019
JAN: http://www.genome.ad.jp/dbget-bin/www_bget?jan+Jann_0168
RDE: http://www.genome.ad.jp/dbget-bin/www_bget?rde+RD1_0147(idi)
DSH: http://www.genome.ad.jp/dbget-bin/www_bget?dsh+Dshi_3527
BSU: http://www.genome.ad.jp/dbget-bin/www_bget?bsu+BG11440(ypgA)
BAN: http://www.genome.ad.jp/dbget-bin/www_bget?ban+BA1520
BAR: http://www.genome.ad.jp/dbget-bin/www_bget?bar+GBAA1520
BAA: http://www.genome.ad.jp/dbget-bin/www_bget?baa+BA_2041
BAT: http://www.genome.ad.jp/dbget-bin/www_bget?bat+BAS1409
BCE: http://www.genome.ad.jp/dbget-bin/www_bget?bce+BC1499
BCA: http://www.genome.ad.jp/dbget-bin/www_bget?bca+BCE_1626
BCZ: http://www.genome.ad.jp/dbget-bin/www_bget?bcz+BCZK1380(fni)
BCY: http://www.genome.ad.jp/dbget-bin/www_bget?bcy+Bcer98_1222
BTK: http://www.genome.ad.jp/dbget-bin/www_bget?btk+BT9727_1381(fni)
BTL: http://www.genome.ad.jp/dbget-bin/www_bget?btl+BALH_1354
BLI: http://www.genome.ad.jp/dbget-bin/www_bget?bli+BL02217(fni)
BLD: http://www.genome.ad.jp/dbget-bin/www_bget?bld+BLi02426
BAY: http://www.genome.ad.jp/dbget-bin/www_bget?bay+RBAM_021020(fni)
BPU: http://www.genome.ad.jp/dbget-bin/www_bget?bpu+BPUM_2020(fni)
OIH: http://www.genome.ad.jp/dbget-bin/www_bget?oih+OB0537
SAU: http://www.genome.ad.jp/dbget-bin/www_bget?sau+SA2136(fni)
SAV: http://www.genome.ad.jp/dbget-bin/www_bget?sav+SAV2346(fni)
SAM: http://www.genome.ad.jp/dbget-bin/www_bget?sam+MW2267(fni)
SAR: http://www.genome.ad.jp/dbget-bin/www_bget?sar+SAR2431(fni)
SAS: http://www.genome.ad.jp/dbget-bin/www_bget?sas+SAS2237
SAC: http://www.genome.ad.jp/dbget-bin/www_bget?sac+SACOL2341(fni)
SAB: http://www.genome.ad.jp/dbget-bin/www_bget?sab+SAB2225c(fni)
SAA: http://www.genome.ad.jp/dbget-bin/www_bget?saa+SAUSA300_2292(fni)
SAO: http://www.genome.ad.jp/dbget-bin/www_bget?sao+SAOUHSC_02623
SEP: http://www.genome.ad.jp/dbget-bin/www_bget?sep+SE1925
SER: http://www.genome.ad.jp/dbget-bin/www_bget?ser+SERP1937(fni-2)
SHA: http://www.genome.ad.jp/dbget-bin/www_bget?sha+SH0712(fni)
SSP: http://www.genome.ad.jp/dbget-bin/www_bget?ssp+SSP0556
LMO: http://www.genome.ad.jp/dbget-bin/www_bget?lmo+lmo1383
LMF: http://www.genome.ad.jp/dbget-bin/www_bget?lmf+LMOf2365_1402(fni)
LIN: http://www.genome.ad.jp/dbget-bin/www_bget?lin+lin1420
LWE: http://www.genome.ad.jp/dbget-bin/www_bget?lwe+lwe1399(fni)
LLA: http://www.genome.ad.jp/dbget-bin/www_bget?lla+L11083(yebB)
LLC: http://www.genome.ad.jp/dbget-bin/www_bget?llc+LACR_0457
LLM: http://www.genome.ad.jp/dbget-bin/www_bget?llm+llmg_0428(fni)
SPY: http://www.genome.ad.jp/dbget-bin/www_bget?spy+SPy_0879
SPZ: http://www.genome.ad.jp/dbget-bin/www_bget?spz+M5005_Spy_0685
SPM: http://www.genome.ad.jp/dbget-bin/www_bget?spm+spyM18_0940
SPG: http://www.genome.ad.jp/dbget-bin/www_bget?spg+SpyM3_0598
SPS: http://www.genome.ad.jp/dbget-bin/www_bget?sps+SPs1255
SPH: http://www.genome.ad.jp/dbget-bin/www_bget?sph+MGAS10270_Spy0743
SPI: http://www.genome.ad.jp/dbget-bin/www_bget?spi+MGAS10750_Spy0777
SPJ: http://www.genome.ad.jp/dbget-bin/www_bget?spj+MGAS2096_Spy0756
SPK: http://www.genome.ad.jp/dbget-bin/www_bget?spk+MGAS9429_Spy0740
SPF: http://www.genome.ad.jp/dbget-bin/www_bget?spf+SpyM51123(fni)
SPA: http://www.genome.ad.jp/dbget-bin/www_bget?spa+M6_Spy0702
SPB: http://www.genome.ad.jp/dbget-bin/www_bget?spb+M28_Spy0665
SPN: http://www.genome.ad.jp/dbget-bin/www_bget?spn+SP_0384
SPR: http://www.genome.ad.jp/dbget-bin/www_bget?spr+spr0341(fni)
SPD: http://www.genome.ad.jp/dbget-bin/www_bget?spd+SPD_0349(fni)
SAG: http://www.genome.ad.jp/dbget-bin/www_bget?sag+SAG1323
SAN: http://www.genome.ad.jp/dbget-bin/www_bget?san+gbs1393
SAK: http://www.genome.ad.jp/dbget-bin/www_bget?sak+SAK_1354(fni)
SMU: http://www.genome.ad.jp/dbget-bin/www_bget?smu+SMU.939
STC: http://www.genome.ad.jp/dbget-bin/www_bget?stc+str0562(idi)
STL: http://www.genome.ad.jp/dbget-bin/www_bget?stl+stu0562(idi)
STE: http://www.genome.ad.jp/dbget-bin/www_bget?ste+STER_0601
SSA: http://www.genome.ad.jp/dbget-bin/www_bget?ssa+SSA_0336
SGO: http://www.genome.ad.jp/dbget-bin/www_bget?sgo+SGO_0242
LPL: http://www.genome.ad.jp/dbget-bin/www_bget?lpl+lp_1732(idi1)
LJO: http://www.genome.ad.jp/dbget-bin/www_bget?ljo+LJ1208
LAC: http://www.genome.ad.jp/dbget-bin/www_bget?lac+LBA1171
LSA: http://www.genome.ad.jp/dbget-bin/www_bget?lsa+LSA0905(idi)
LSL: http://www.genome.ad.jp/dbget-bin/www_bget?lsl+LSL_0682
LDB: http://www.genome.ad.jp/dbget-bin/www_bget?ldb+Ldb0996(fni)
LBU: http://www.genome.ad.jp/dbget-bin/www_bget?lbu+LBUL_0903
LBR: http://www.genome.ad.jp/dbget-bin/www_bget?lbr+LVIS_0861
LCA: http://www.genome.ad.jp/dbget-bin/www_bget?lca+LSEI_1493
LGA: http://www.genome.ad.jp/dbget-bin/www_bget?lga+LGAS_1036
LRE: http://www.genome.ad.jp/dbget-bin/www_bget?lre+Lreu_0912
EFA: http://www.genome.ad.jp/dbget-bin/www_bget?efa+EF0901
OOE: http://www.genome.ad.jp/dbget-bin/www_bget?ooe+OEOE_1103
STH: http://www.genome.ad.jp/dbget-bin/www_bget?sth+STH1674
CBE: http://www.genome.ad.jp/dbget-bin/www_bget?cbe+Cbei_3081
DRM: http://www.genome.ad.jp/dbget-bin/www_bget?drm+Dred_0474
SWO: http://www.genome.ad.jp/dbget-bin/www_bget?swo+Swol_1341
MTA: http://www.genome.ad.jp/dbget-bin/www_bget?mta+Moth_1328
MTU: http://www.genome.ad.jp/dbget-bin/www_bget?mtu+Rv1745c(idi)
MTC: http://www.genome.ad.jp/dbget-bin/www_bget?mtc+MT1787(idi)
MBO: http://www.genome.ad.jp/dbget-bin/www_bget?mbo+Mb1774c(idi)
MBB: http://www.genome.ad.jp/dbget-bin/www_bget?mbb+BCG_1784c(idi)
MPA: http://www.genome.ad.jp/dbget-bin/www_bget?mpa+MAP3079c
MAV: http://www.genome.ad.jp/dbget-bin/www_bget?mav+MAV_3894(fni)
MSM: http://www.genome.ad.jp/dbget-bin/www_bget?msm+MSMEG_1057(fni) http://www.genome.ad.jp/dbget-bin/www_bget?msm+MSMEG_2337(fni)
MUL: http://www.genome.ad.jp/dbget-bin/www_bget?mul+MUL_0380(idi2)
MVA: http://www.genome.ad.jp/dbget-bin/www_bget?mva+Mvan_1582 http://www.genome.ad.jp/dbget-bin/www_bget?mva+Mvan_2176
MGI: http://www.genome.ad.jp/dbget-bin/www_bget?mgi+Mflv_1842 http://www.genome.ad.jp/dbget-bin/www_bget?mgi+Mflv_4187
MMC: http://www.genome.ad.jp/dbget-bin/www_bget?mmc+Mmcs_1954
MKM: http://www.genome.ad.jp/dbget-bin/www_bget?mkm+Mkms_2000
MJL: http://www.genome.ad.jp/dbget-bin/www_bget?mjl+Mjls_1934
CGL: http://www.genome.ad.jp/dbget-bin/www_bget?cgl+NCgl2223(cgl2305)
CGB: http://www.genome.ad.jp/dbget-bin/www_bget?cgb+cg2531(idi)
CEF: http://www.genome.ad.jp/dbget-bin/www_bget?cef+CE2207
CDI: http://www.genome.ad.jp/dbget-bin/www_bget?cdi+DIP1730(idi)
NFA: http://www.genome.ad.jp/dbget-bin/www_bget?nfa+nfa19790 http://www.genome.ad.jp/dbget-bin/www_bget?nfa+nfa22100
RHA: http://www.genome.ad.jp/dbget-bin/www_bget?rha+RHA1_ro00239
SCO: http://www.genome.ad.jp/dbget-bin/www_bget?sco+SCO6750(SC5F2A.33c)
SMA: http://www.genome.ad.jp/dbget-bin/www_bget?sma+SAV1663(idi)
LXX: http://www.genome.ad.jp/dbget-bin/www_bget?lxx+Lxx23810(idi)
CMI: http://www.genome.ad.jp/dbget-bin/www_bget?cmi+CMM_2889(idiA)
AAU: http://www.genome.ad.jp/dbget-bin/www_bget?aau+AAur_0321(idi)
PAC: http://www.genome.ad.jp/dbget-bin/www_bget?pac+PPA2115
FRA: http://www.genome.ad.jp/dbget-bin/www_bget?fra+Francci3_4188
FRE: http://www.genome.ad.jp/dbget-bin/www_bget?fre+Franean1_5570
FAL: http://www.genome.ad.jp/dbget-bin/www_bget?fal+FRAAL6504(idi)
KRA: http://www.genome.ad.jp/dbget-bin/www_bget?kra+Krad_3991
SEN: http://www.genome.ad.jp/dbget-bin/www_bget?sen+SACE_2627(idiB_2) http://www.genome.ad.jp/dbget-bin/www_bget?sen+SACE_5210(idi)
STP: http://www.genome.ad.jp/dbget-bin/www_bget?stp+Strop_4438
SAQ: http://www.genome.ad.jp/dbget-bin/www_bget?saq+Sare_4564 http://www.genome.ad.jp/dbget-bin/www_bget?saq+Sare_4928
RXY: http://www.genome.ad.jp/dbget-bin/www_bget?rxy+Rxyl_0400
BBU: http://www.genome.ad.jp/dbget-bin/www_bget?bbu+BB0684
BGA: http://www.genome.ad.jp/dbget-bin/www_bget?bga+BG0707
SYN: http://www.genome.ad.jp/dbget-bin/www_bget?syn+sll1556
SYC: http://www.genome.ad.jp/dbget-bin/www_bget?syc+syc2161_c
SYF: http://www.genome.ad.jp/dbget-bin/www_bget?syf+Synpcc7942_1933
CYA: http://www.genome.ad.jp/dbget-bin/www_bget?cya+CYA_2395(fni)
CYB: http://www.genome.ad.jp/dbget-bin/www_bget?cyb+CYB_2691(fni)
TEL: http://www.genome.ad.jp/dbget-bin/www_bget?tel+tll1403
ANA: http://www.genome.ad.jp/dbget-bin/www_bget?ana+all4591
AVA: http://www.genome.ad.jp/dbget-bin/www_bget?ava+Ava_2461 http://www.genome.ad.jp/dbget-bin/www_bget?ava+Ava_B0346
TER: http://www.genome.ad.jp/dbget-bin/www_bget?ter+Tery_1589
SRU: http://www.genome.ad.jp/dbget-bin/www_bget?sru+SRU_1900(idi)
CHU: http://www.genome.ad.jp/dbget-bin/www_bget?chu+CHU_0674(idi)
GFO: http://www.genome.ad.jp/dbget-bin/www_bget?gfo+GFO_2363(idi)
FJO: http://www.genome.ad.jp/dbget-bin/www_bget?fjo+Fjoh_0269
FPS: http://www.genome.ad.jp/dbget-bin/www_bget?fps+FP1792(idi)
CTE: http://www.genome.ad.jp/dbget-bin/www_bget?cte+CT0257
CCH: http://www.genome.ad.jp/dbget-bin/www_bget?cch+Cag_1445
CPH: http://www.genome.ad.jp/dbget-bin/www_bget?cph+Cpha266_0385
PVI: http://www.genome.ad.jp/dbget-bin/www_bget?pvi+Cvib_1545
PLT: http://www.genome.ad.jp/dbget-bin/www_bget?plt+Plut_1764
RRS: http://www.genome.ad.jp/dbget-bin/www_bget?rrs+RoseRS_2437
RCA: http://www.genome.ad.jp/dbget-bin/www_bget?rca+Rcas_2215
HAU: http://www.genome.ad.jp/dbget-bin/www_bget?hau+Haur_4687
DRA: http://www.genome.ad.jp/dbget-bin/www_bget?dra+DR_1087
DGE: http://www.genome.ad.jp/dbget-bin/www_bget?dge+Dgeo_1381
TTH: http://www.genome.ad.jp/dbget-bin/www_bget?tth+TT_P0067
TTJ: http://www.genome.ad.jp/dbget-bin/www_bget?ttj+TTHB110
MJA: http://www.genome.ad.jp/dbget-bin/www_bget?mja+MJ0862
MMP: http://www.genome.ad.jp/dbget-bin/www_bget?mmp+MMP0043
MMQ: http://www.genome.ad.jp/dbget-bin/www_bget?mmq+MmarC5_1637
MMX: http://www.genome.ad.jp/dbget-bin/www_bget?mmx+MmarC6_0906
MMZ: http://www.genome.ad.jp/dbget-bin/www_bget?mmz+MmarC7_1040
MAE: http://www.genome.ad.jp/dbget-bin/www_bget?mae+Maeo_1184
MVN: http://www.genome.ad.jp/dbget-bin/www_bget?mvn+Mevan_1058
MAC: http://www.genome.ad.jp/dbget-bin/www_bget?mac+MA0604(idi)
MBA: http://www.genome.ad.jp/dbget-bin/www_bget?mba+Mbar_A1419
MMA: http://www.genome.ad.jp/dbget-bin/www_bget?mma+MM_1764
MBU: http://www.genome.ad.jp/dbget-bin/www_bget?mbu+Mbur_2397
MTP: http://www.genome.ad.jp/dbget-bin/www_bget?mtp+Mthe_0474
MHU: http://www.genome.ad.jp/dbget-bin/www_bget?mhu+Mhun_2888
MLA: http://www.genome.ad.jp/dbget-bin/www_bget?mla+Mlab_1665
MEM: http://www.genome.ad.jp/dbget-bin/www_bget?mem+Memar_1814
MBN: http://www.genome.ad.jp/dbget-bin/www_bget?mbn+Mboo_2211
MTH: http://www.genome.ad.jp/dbget-bin/www_bget?mth+MTH48
MST: http://www.genome.ad.jp/dbget-bin/www_bget?mst+Msp_0856(fni)
MSI: http://www.genome.ad.jp/dbget-bin/www_bget?msi+Msm_1441
MKA: http://www.genome.ad.jp/dbget-bin/www_bget?mka+MK0776(lldD)
AFU: http://www.genome.ad.jp/dbget-bin/www_bget?afu+AF2287
HAL: http://www.genome.ad.jp/dbget-bin/www_bget?hal+VNG1818G(idi) http://www.genome.ad.jp/dbget-bin/www_bget?hal+VNG6081G(crt_1) http://www.genome.ad.jp/dbget-bin/www_bget?hal+VNG6445G(crt_2) http://www.genome.ad.jp/dbget-bin/www_bget?hal+VNG7060 http://www.genome.ad.jp/dbget-bin/www_bget?hal+VNG7149
HMA: http://www.genome.ad.jp/dbget-bin/www_bget?hma+rrnAC3484(idi)
HWA: http://www.genome.ad.jp/dbget-bin/www_bget?hwa+HQ2772A(idiA) http://www.genome.ad.jp/dbget-bin/www_bget?hwa+HQ2847A(idiB)
NPH: http://www.genome.ad.jp/dbget-bin/www_bget?nph+NP0360A(idiB_1) http://www.genome.ad.jp/dbget-bin/www_bget?nph+NP4826A(idiA) http://www.genome.ad.jp/dbget-bin/www_bget?nph+NP5124A(idiB_2)
TAC: http://www.genome.ad.jp/dbget-bin/www_bget?tac+Ta0102
TVO: http://www.genome.ad.jp/dbget-bin/www_bget?tvo+TVN0179
PTO: http://www.genome.ad.jp/dbget-bin/www_bget?pto+PTO0496
PHO: http://www.genome.ad.jp/dbget-bin/www_bget?pho+PH1202
PAB: http://www.genome.ad.jp/dbget-bin/www_bget?pab+PAB1662
PFU: http://www.genome.ad.jp/dbget-bin/www_bget?pfu+PF0856
TKO: http://www.genome.ad.jp/dbget-bin/www_bget?tko+TK1470
RCI: http://www.genome.ad.jp/dbget-bin/www_bget?rci+LRC397(fni)
APE: http://www.genome.ad.jp/dbget-bin/www_bget?ape+APE_1765.1
SMR: http://www.genome.ad.jp/dbget-bin/www_bget?smr+Smar_0822
IHO: http://www.genome.ad.jp/dbget-bin/www_bget?iho+Igni_0804
HBU: http://www.genome.ad.jp/dbget-bin/www_bget?hbu+Hbut_0539
SSO: http://www.genome.ad.jp/dbget-bin/www_bget?sso+SSO0063
STO: http://www.genome.ad.jp/dbget-bin/www_bget?sto+ST2059
SAI: http://www.genome.ad.jp/dbget-bin/www_bget?sai+Saci_0091
MSE: http://www.genome.ad.jp/dbget-bin/www_bget?mse+Msed_2136
PAI: http://www.genome.ad.jp/dbget-bin/www_bget?pai+PAE0801
PIS: http://www.genome.ad.jp/dbget-bin/www_bget?pis+Pisl_1093
PCL: http://www.genome.ad.jp/dbget-bin/www_bget?pcl+Pcal_0017
PAS:
TPE:
Репрезентативные нуклеиновые кислоты и полипептиды изопренсинтазы
GenBank рег. №
AY341431
AY316691
AY279379
AJ457070
AY182241
| название | год | авторы | номер документа |
|---|---|---|---|
| ВАРИАНТЫ ИЗОПРЕНСИНТАЗЫ, ПРИМЕНЯЕМЫЕ ДЛЯ УЛУЧШЕНИЯ ПРОДУЦИРОВАНИЯ ИЗОПРЕНА МИКРООРГАНИЗМАМИ | 2009 |
|
RU2516343C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ПОЛУЧЕНИЯ ИЗОПРЕНА | 2008 |
|
RU2545699C2 |
| ТОПЛИВНЫЕ КОМПОЗИЦИИ, СОДЕРЖАЩИЕ ПРОИЗВОДНЫЕ ИЗОПРЕНА | 2010 |
|
RU2531623C2 |
| ИЗОПРЕНСИНТАЗА И КОДИРУЮЩИЙ ЕЕ ГЕН, СПОСОБ ПОЛУЧЕНИЯ МОНОМЕРА ИЗОПРЕНА | 2013 |
|
RU2612916C2 |
| БАКТЕРИЯ Е.coli, ПРОДУЦИРУЮЩАЯ ГЕТЕРОЛОГИЧНЫЙ ПОЛИПЕПТИД, И СПОСОБ ПРОДУЦИРОВАНИЯ ПОЛИПЕПТИДА | 2001 |
|
RU2287574C2 |
| УЛУЧШЕННАЯ ПРОДУКЦИЯ БЕЛКА В BACILLUS | 2008 |
|
RU2515112C2 |
| СПОСОБ ПОЛУЧЕНИЯ ГИАЛУРОНАНА В РЕКОМБИНАНТНОЙ КЛЕТКЕ-ХОЗЯИНЕ | 2002 |
|
RU2346049C2 |
| СПОСОБЫ И ВЕЩЕСТВА ДЛЯ ОСНОВАННОГО НА РЕКОМБИНАЦИИ ПОЛУЧЕНИЯ СОЕДИНЕНИЙ ШАФРАНА | 2012 |
|
RU2676730C2 |
| ПОЛИПЕПТИДЫ ДЛЯ ИНДУКЦИИ ЗАЩИТНОГО ИММУННОГО ОТВЕТА ПРОТИВ STAPHYLOCOCCUS AUREUS | 2004 |
|
RU2337108C2 |
| РЕКОМБИНАНТНЫЕ МИКРООРГАНИЗМЫ, ПРОДУЦИРУЮЩИЕ МЕТИОНИН | 2006 |
|
RU2447146C2 |















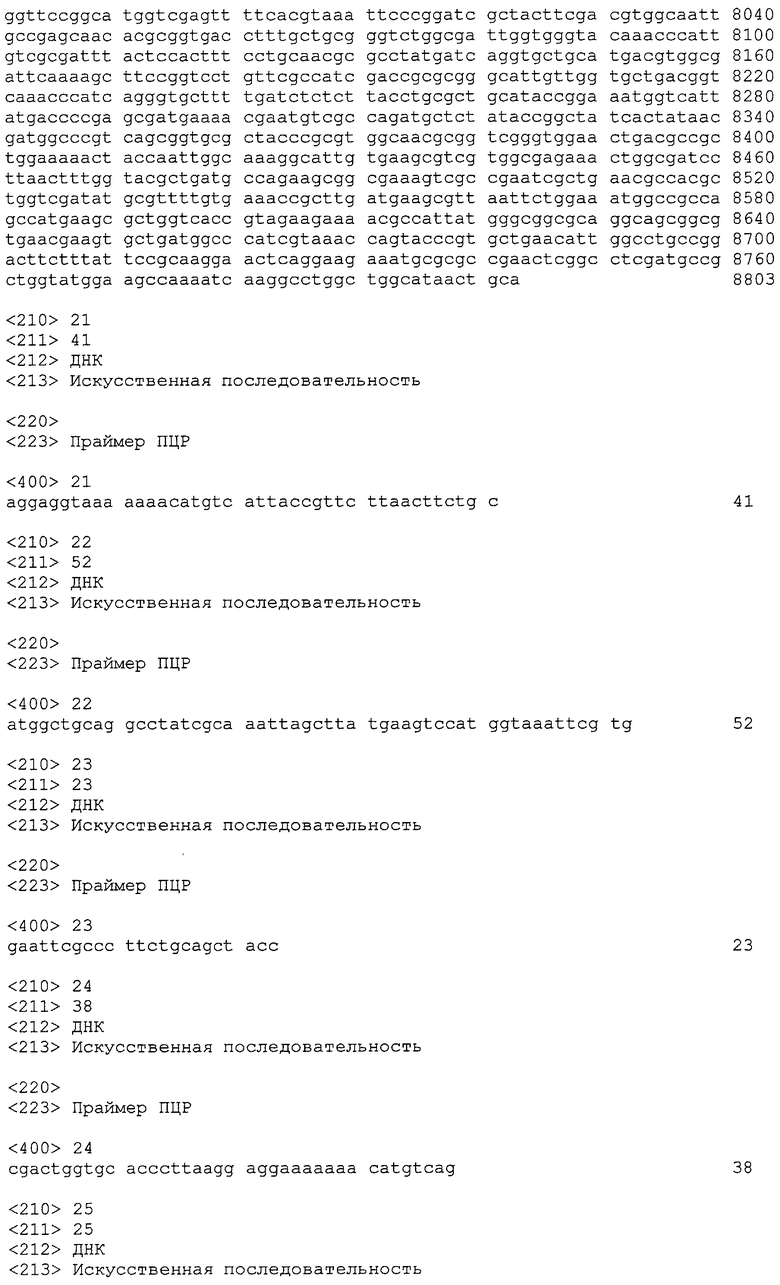
































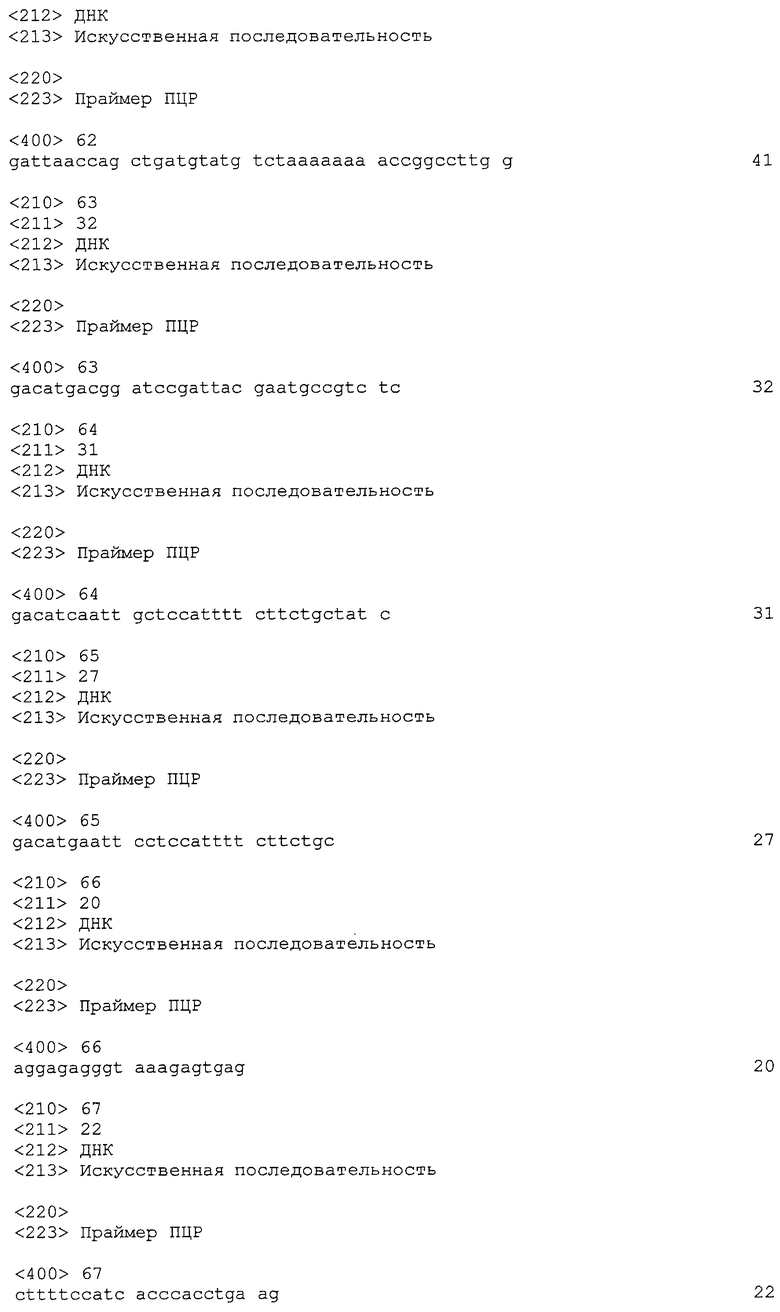

























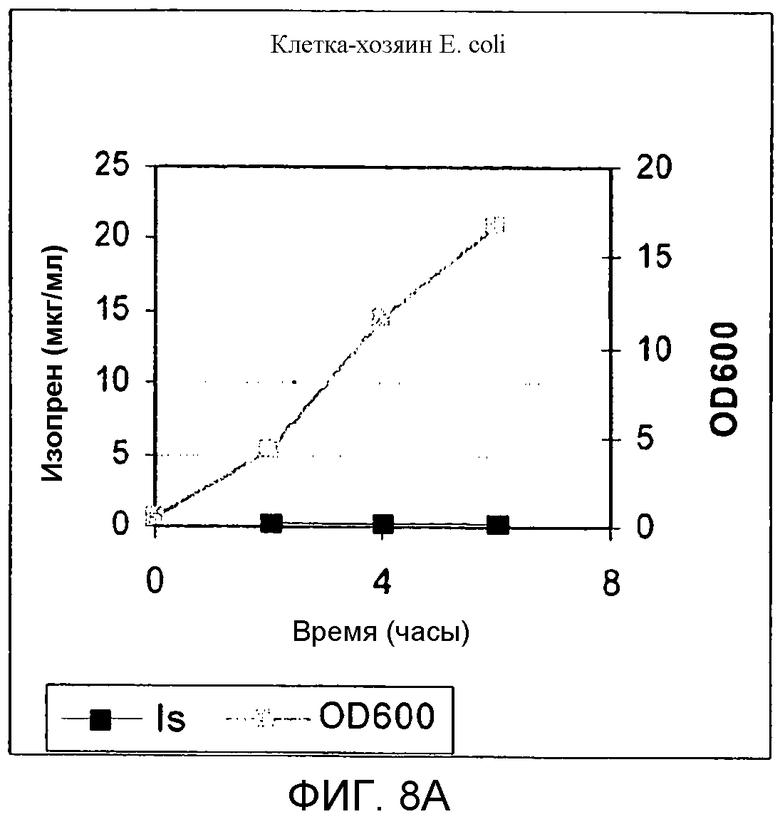
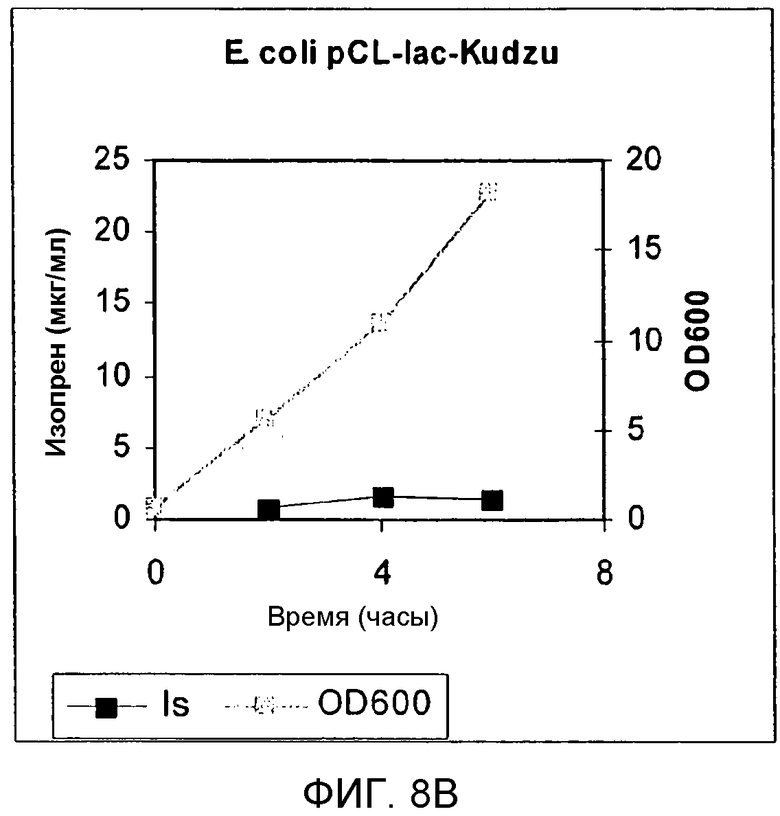
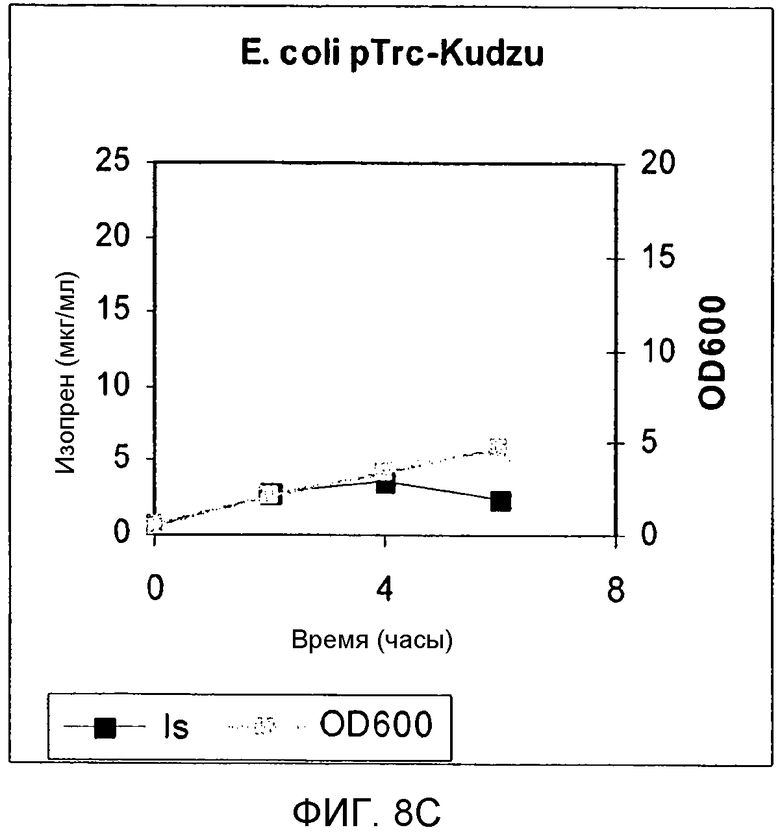
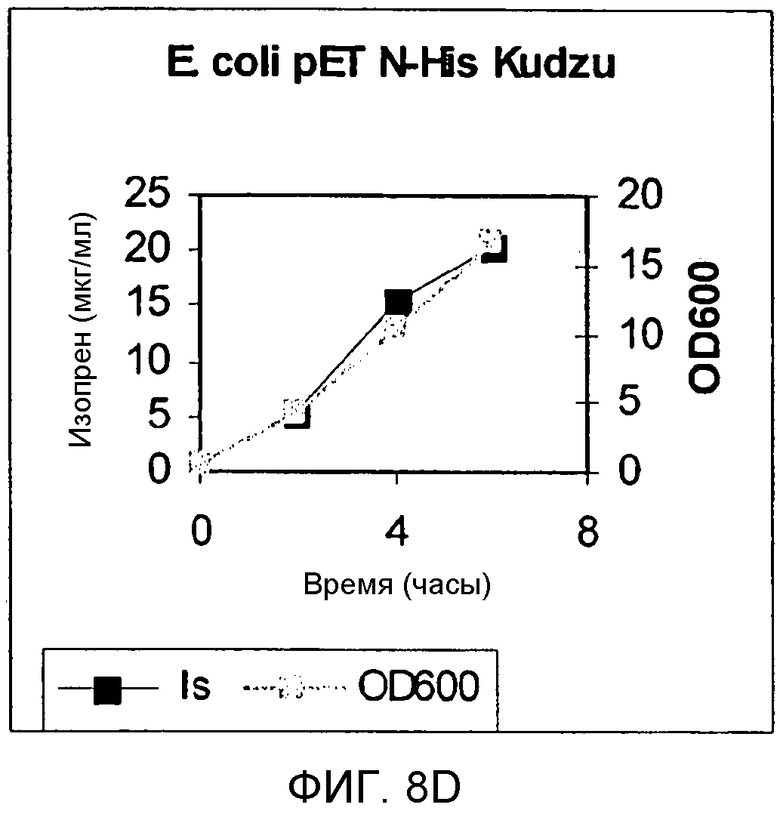
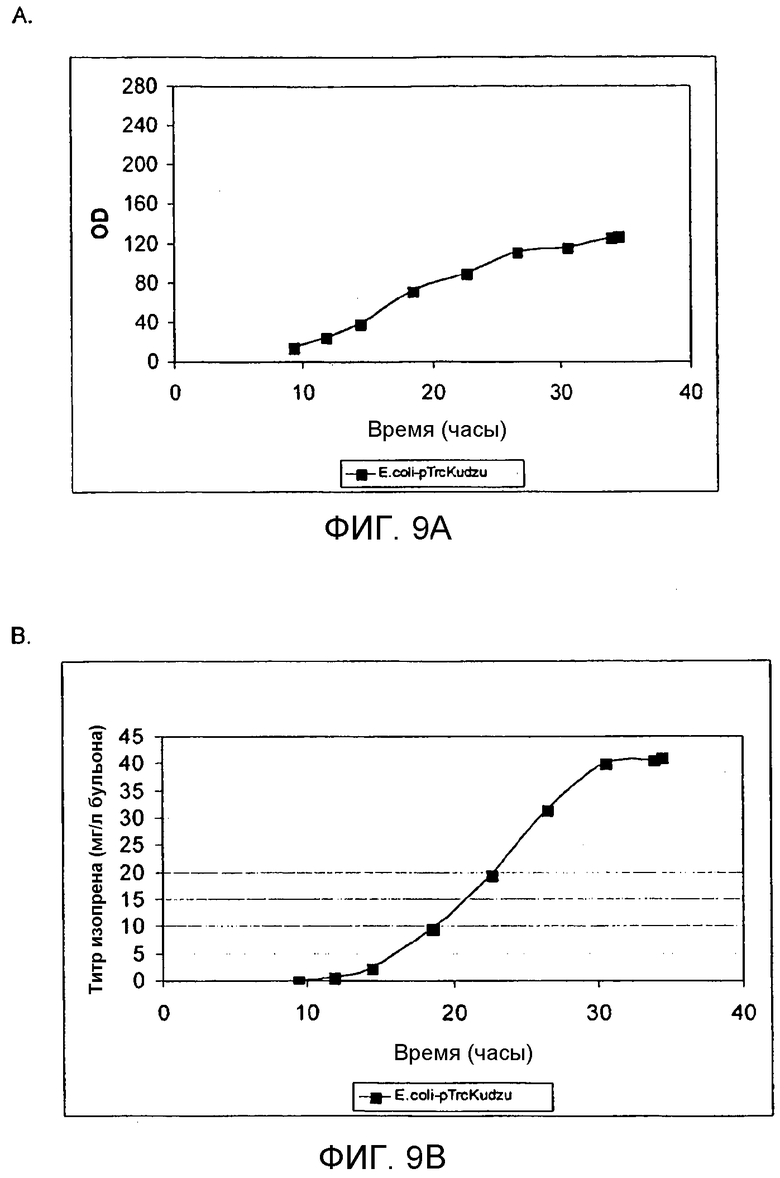
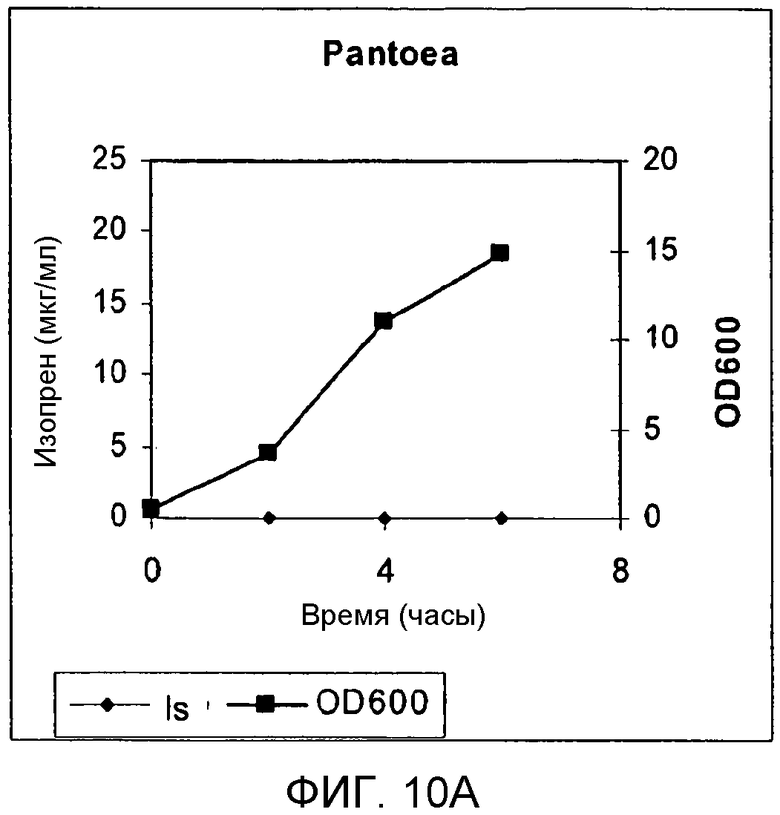
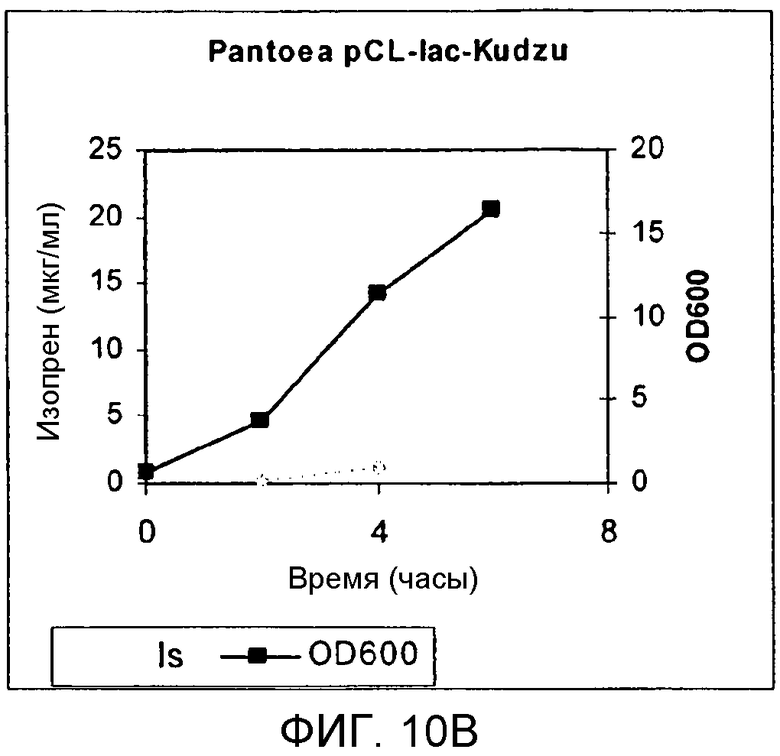
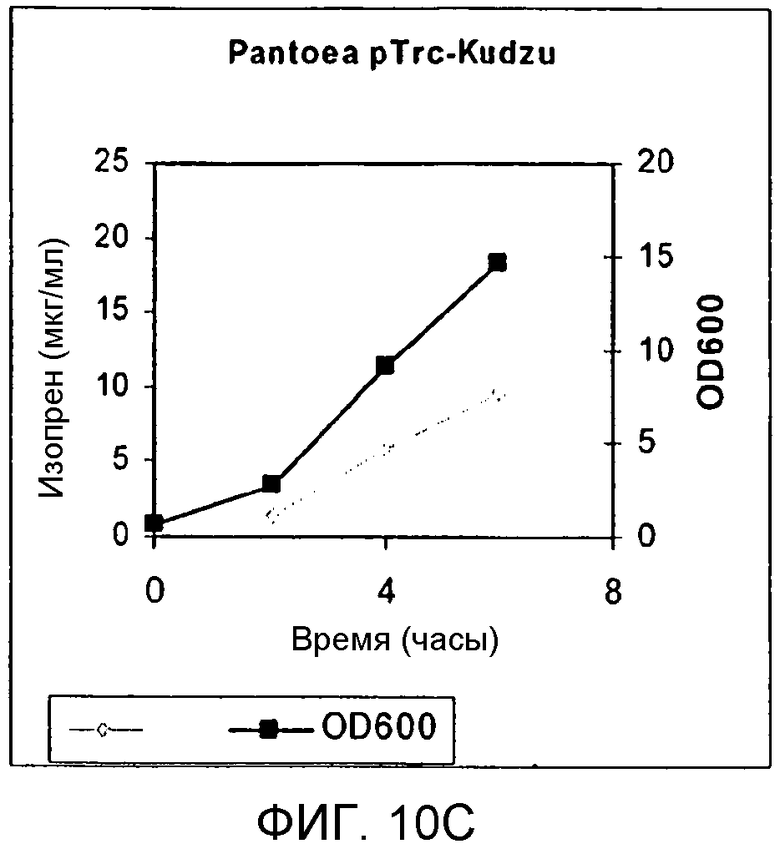
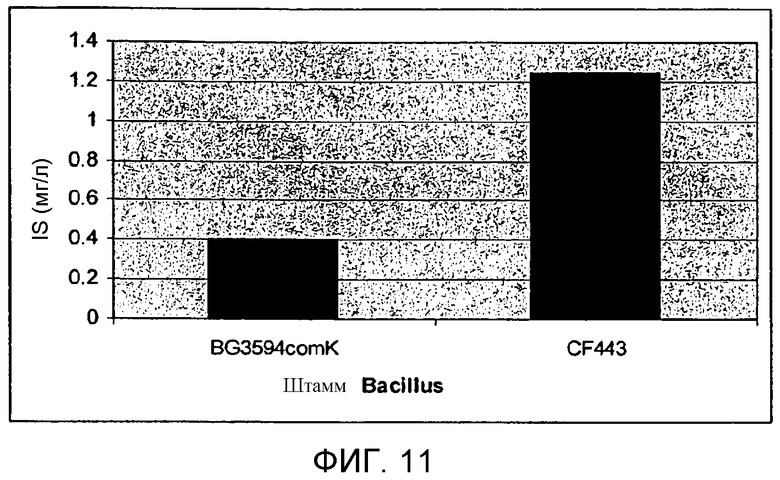




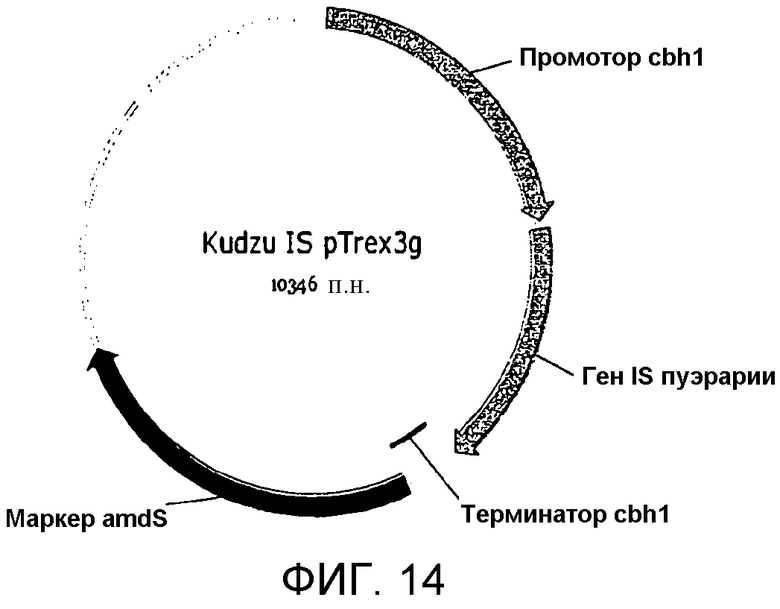



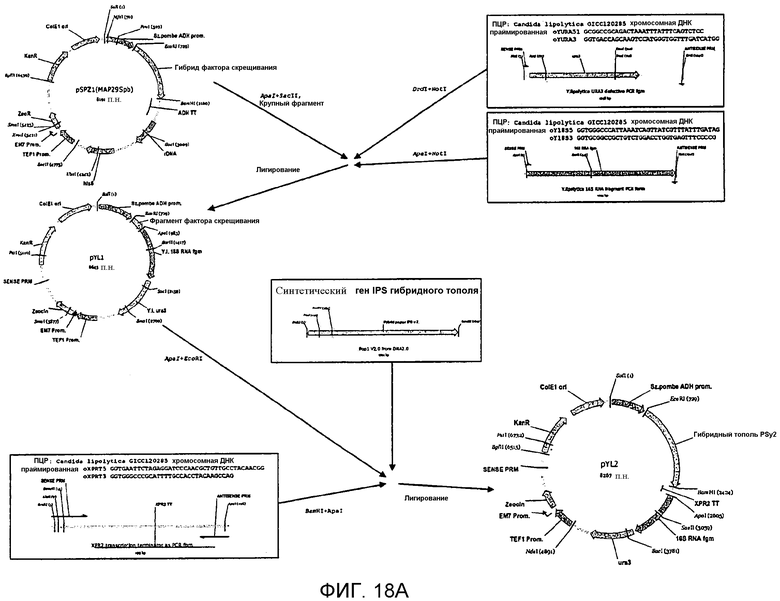
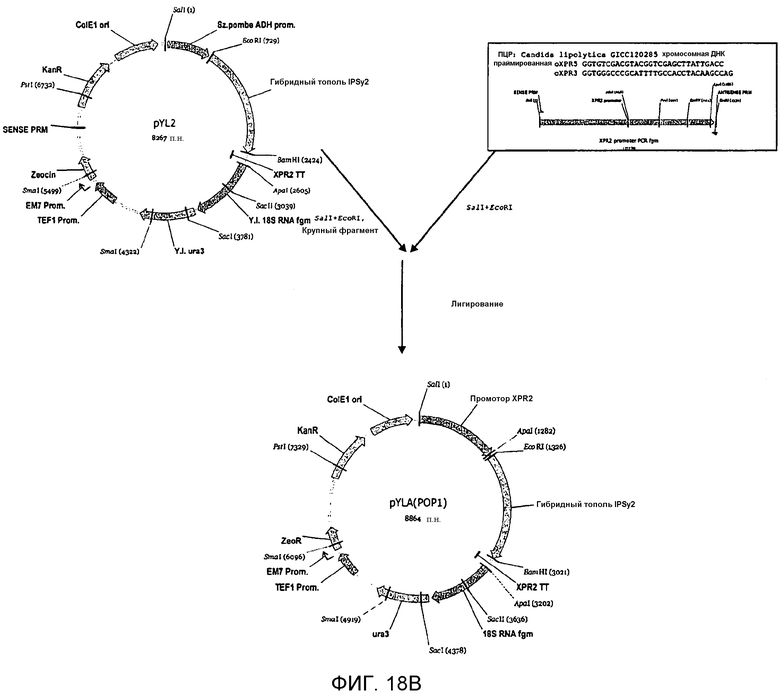
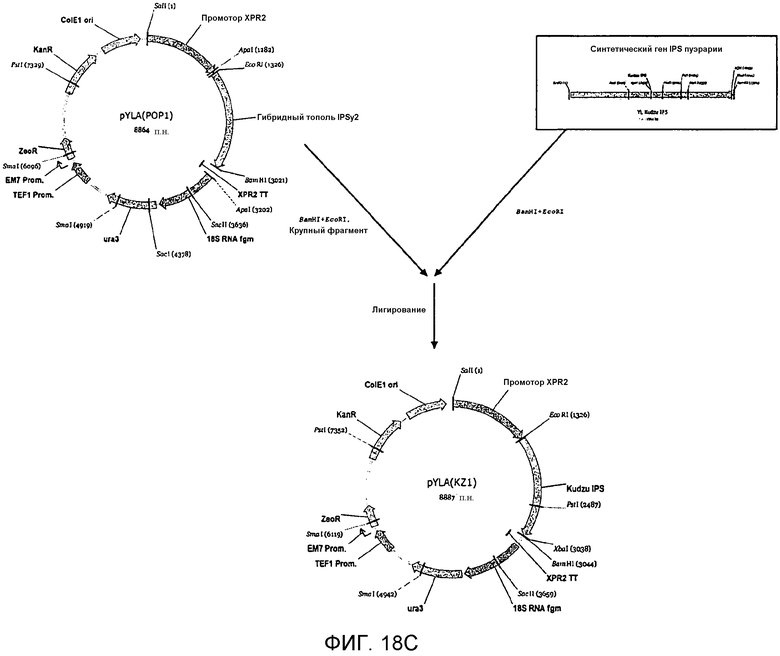
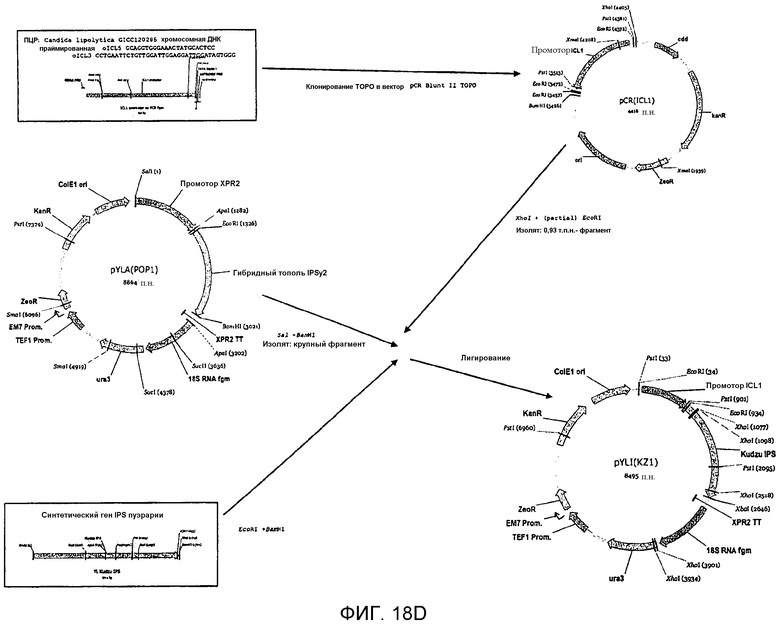
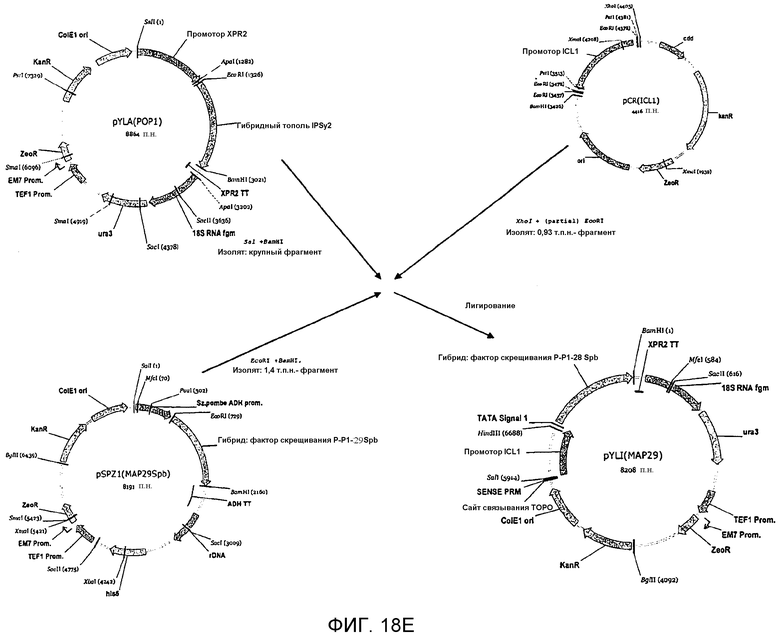
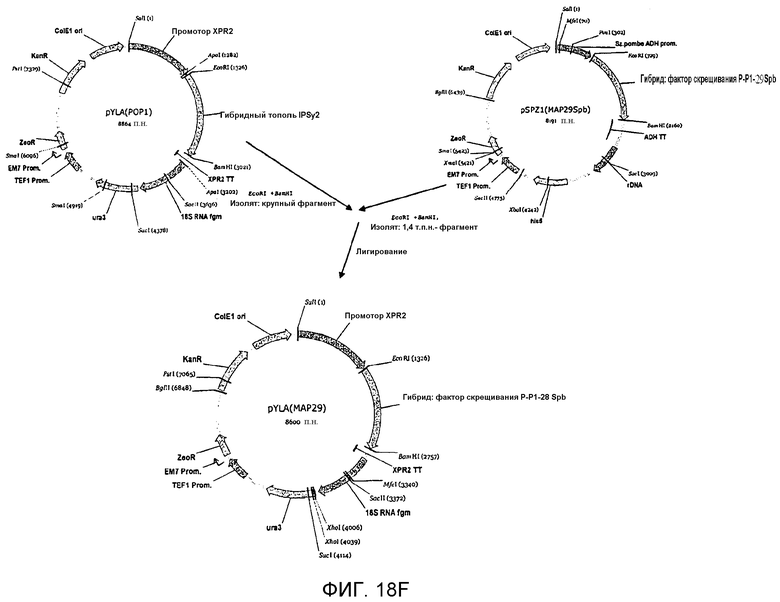
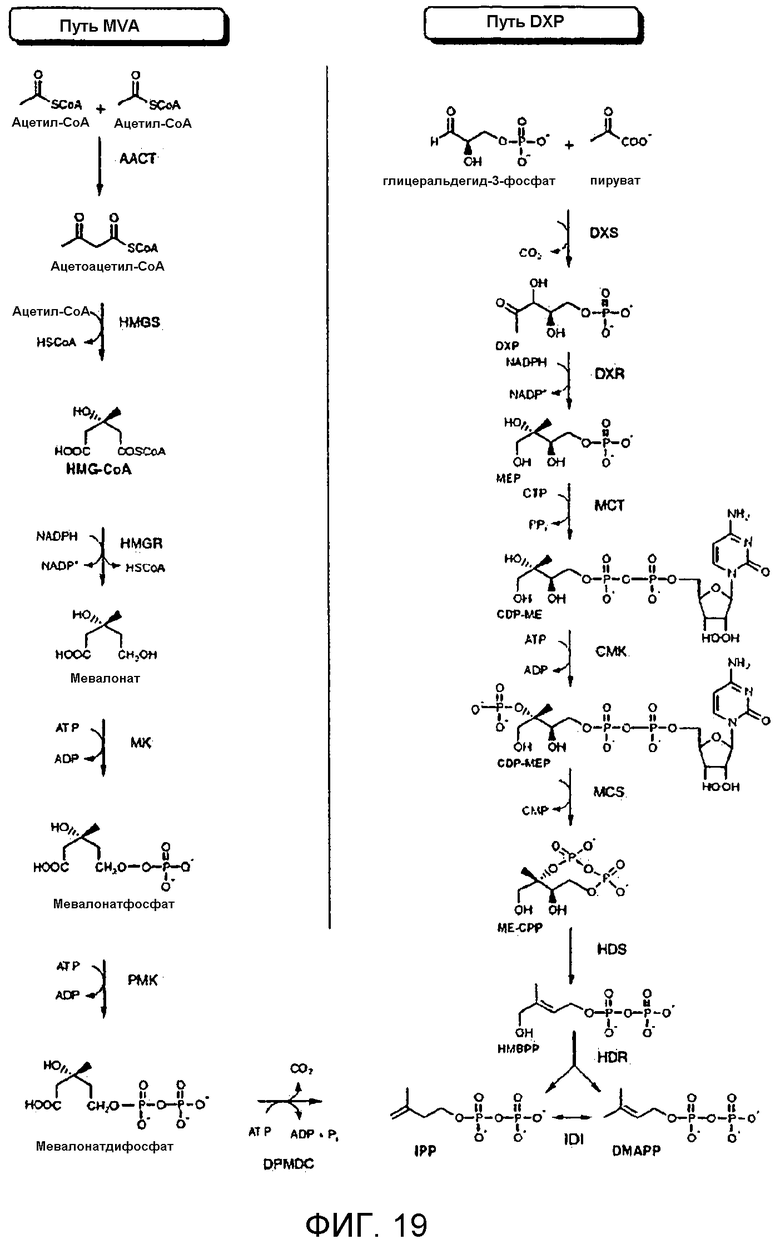
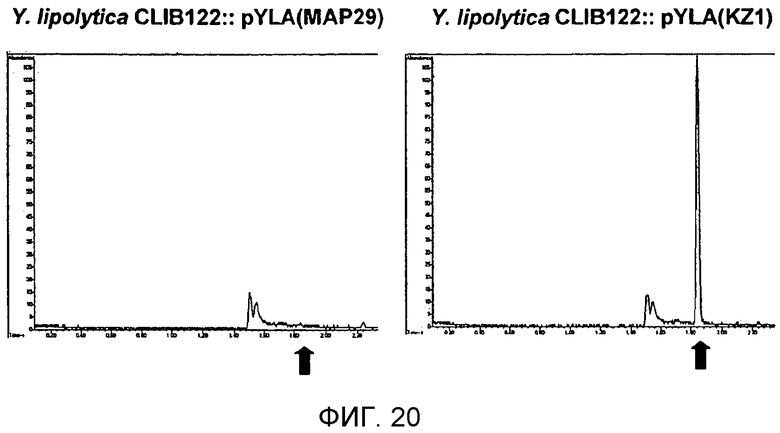
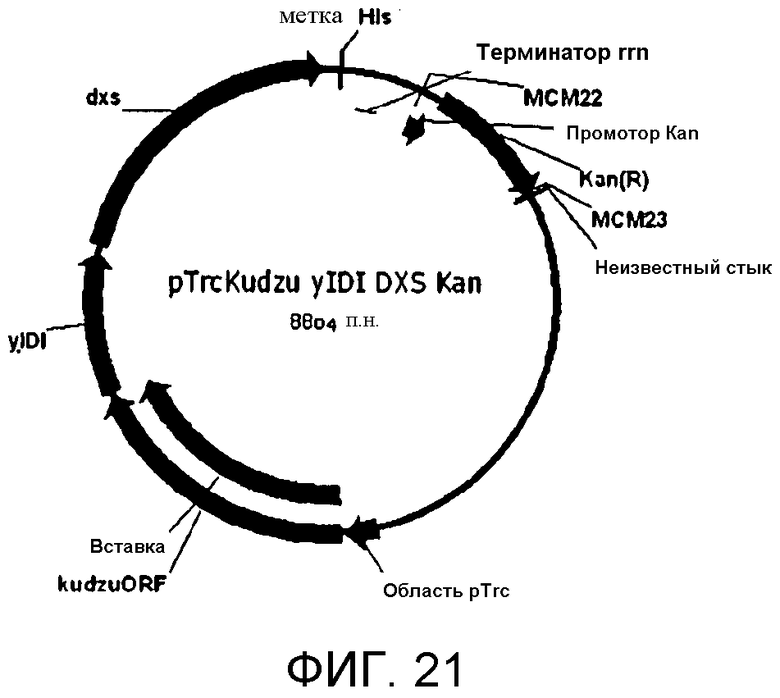




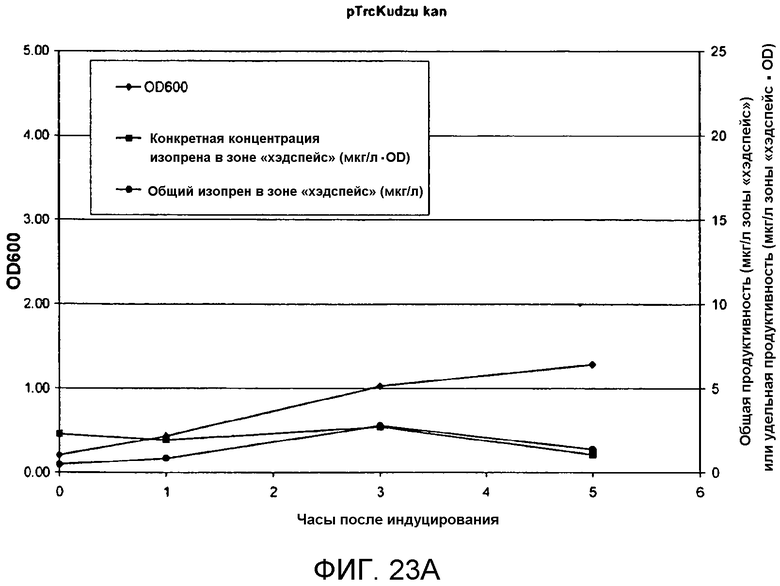
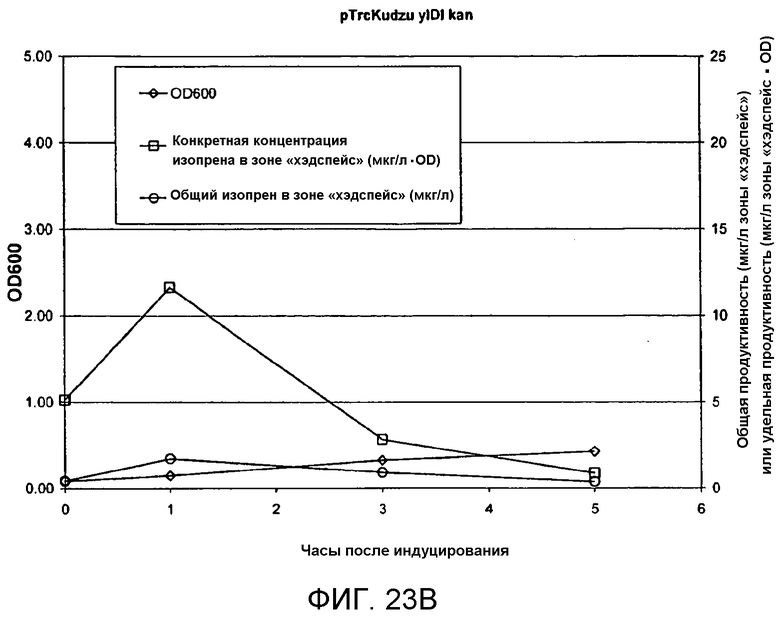
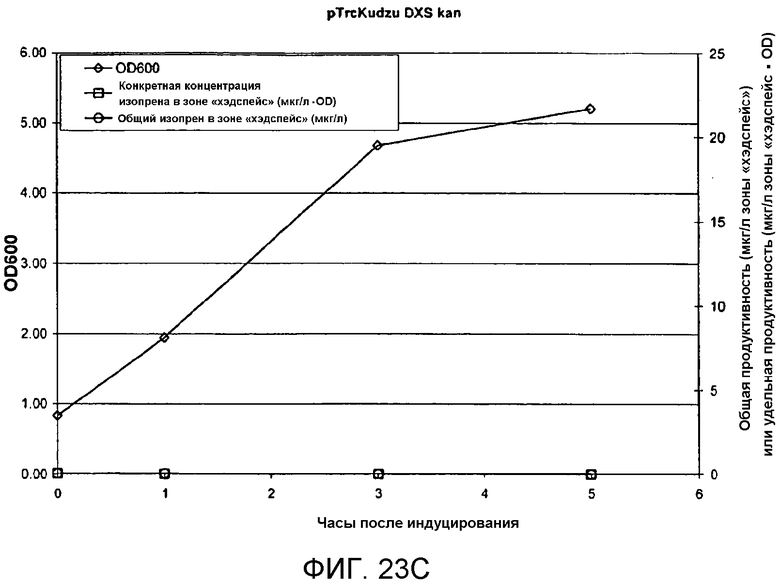
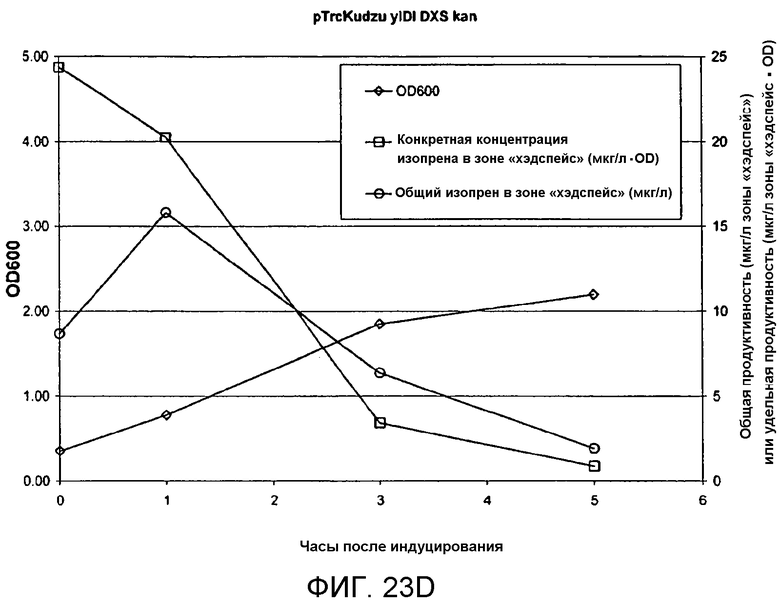
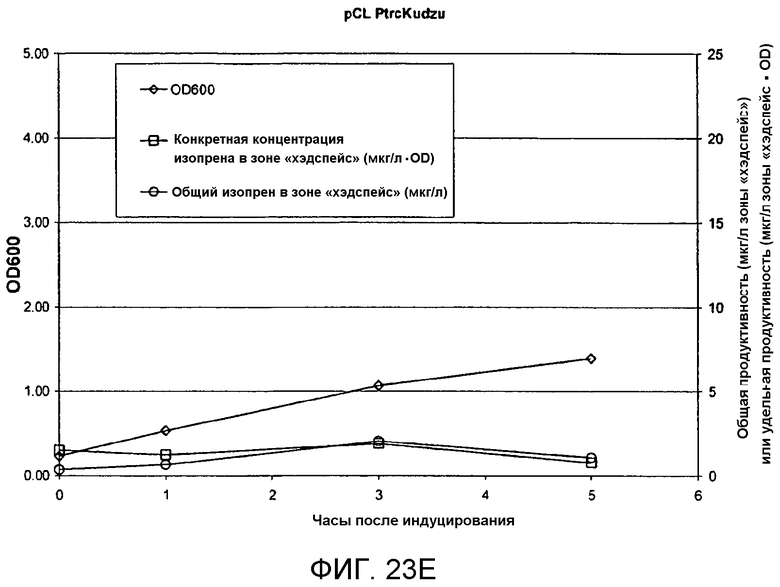
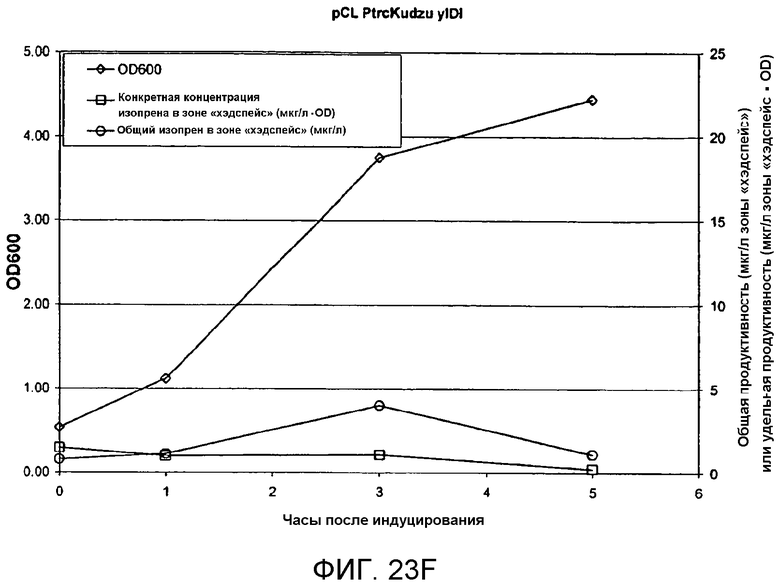
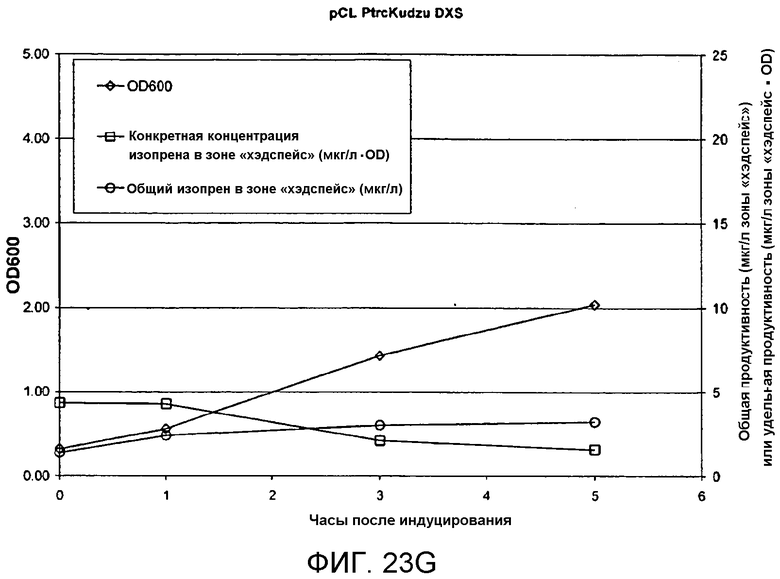
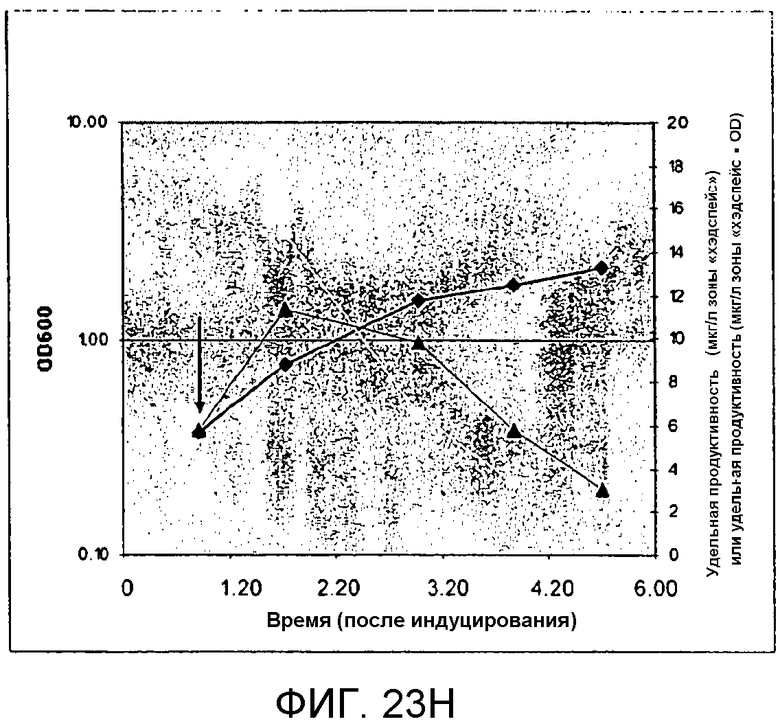
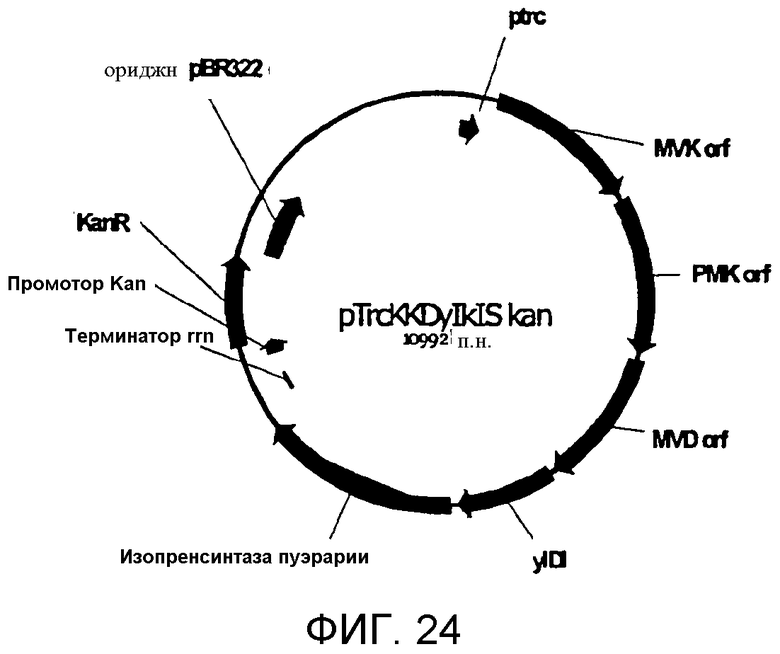




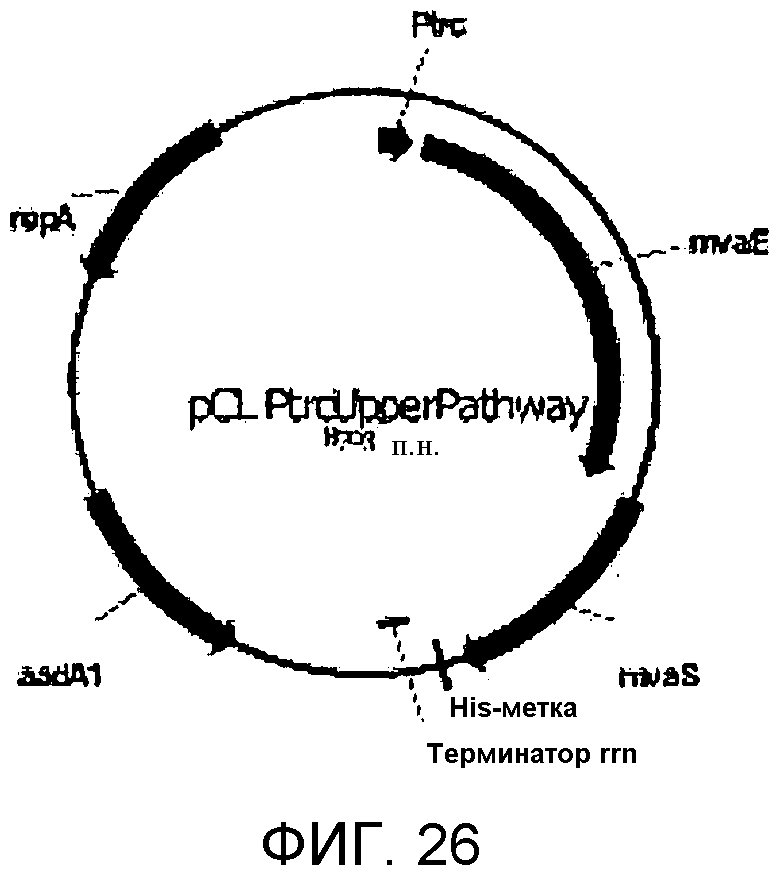




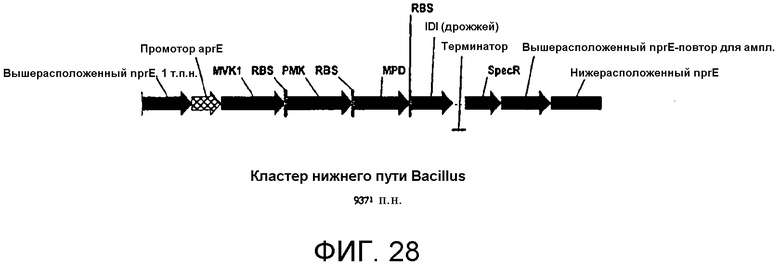




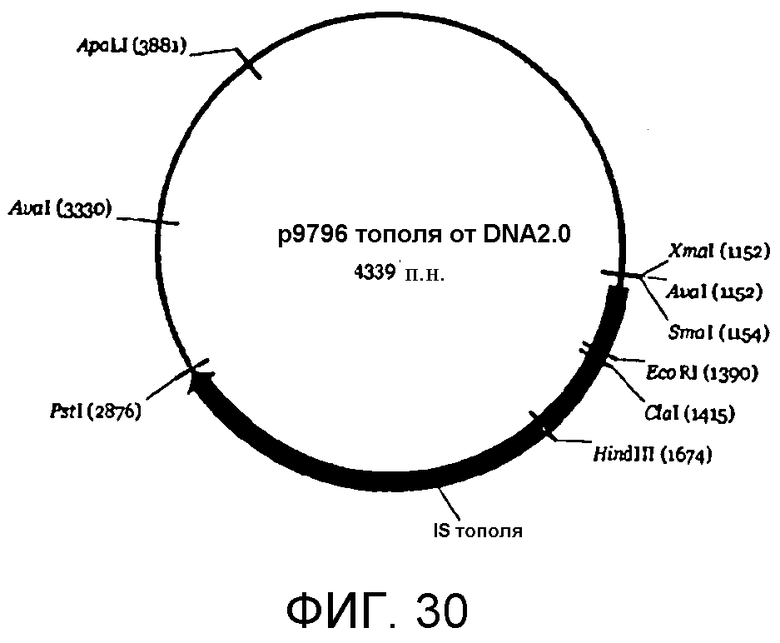

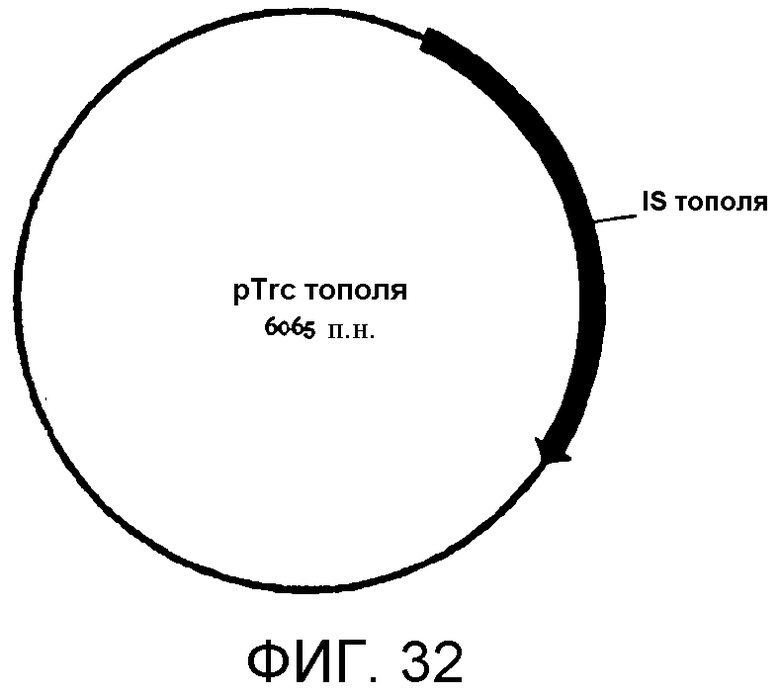



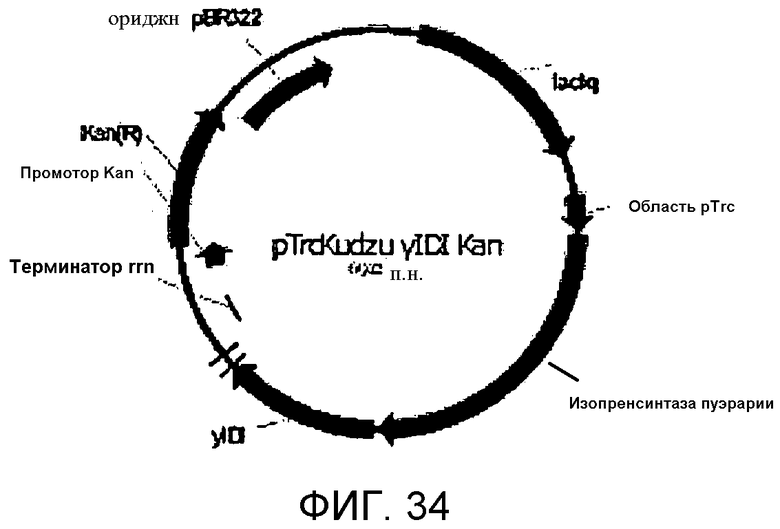


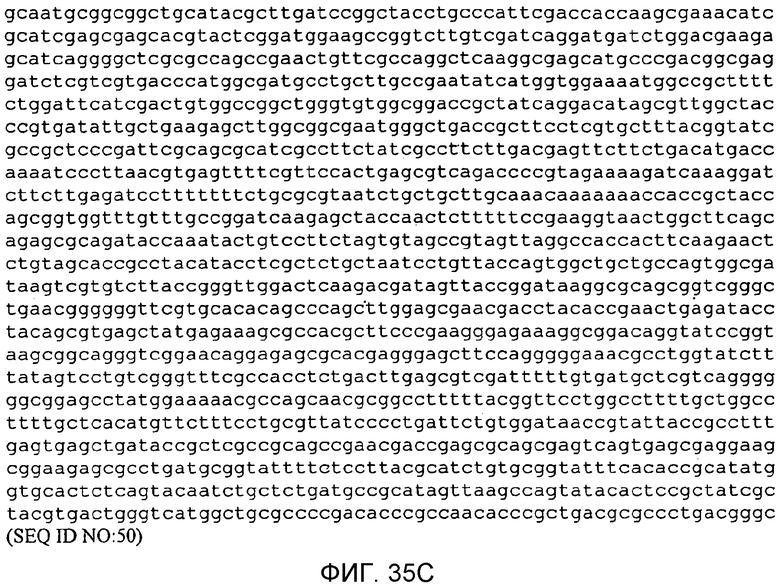
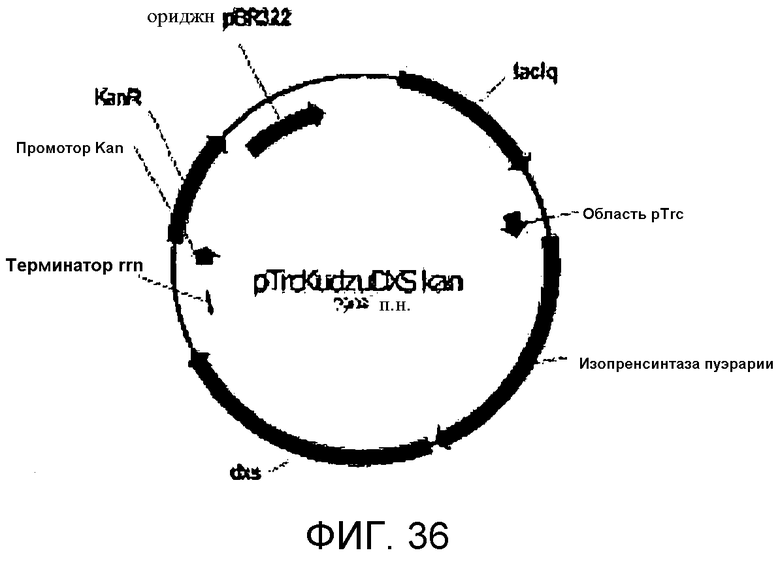



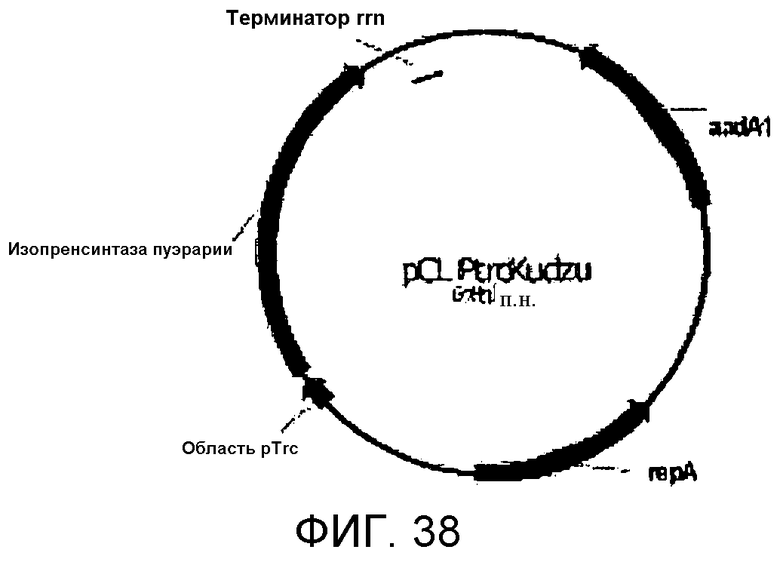



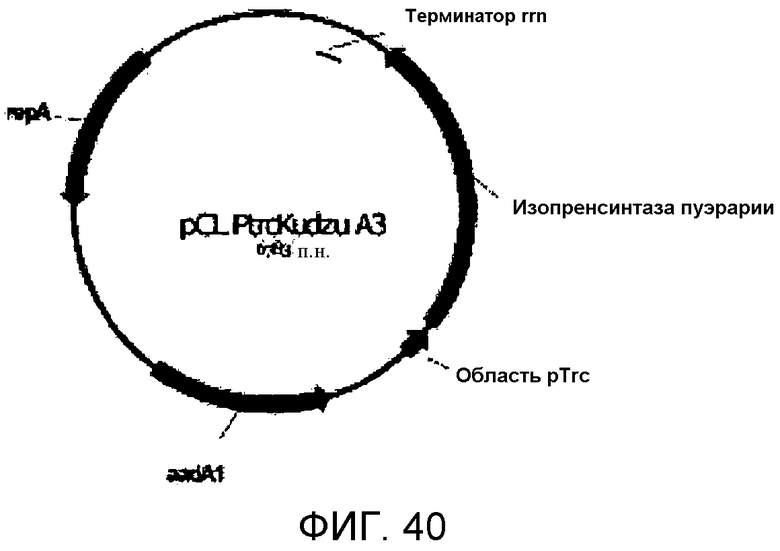



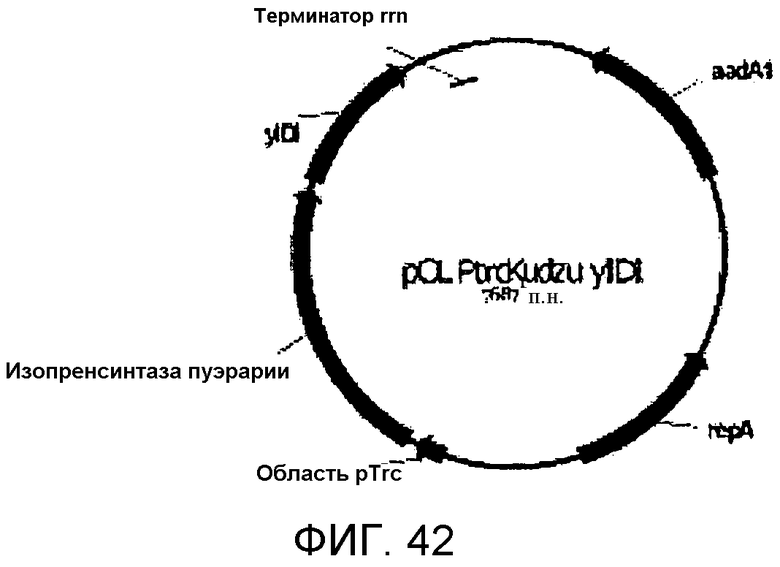



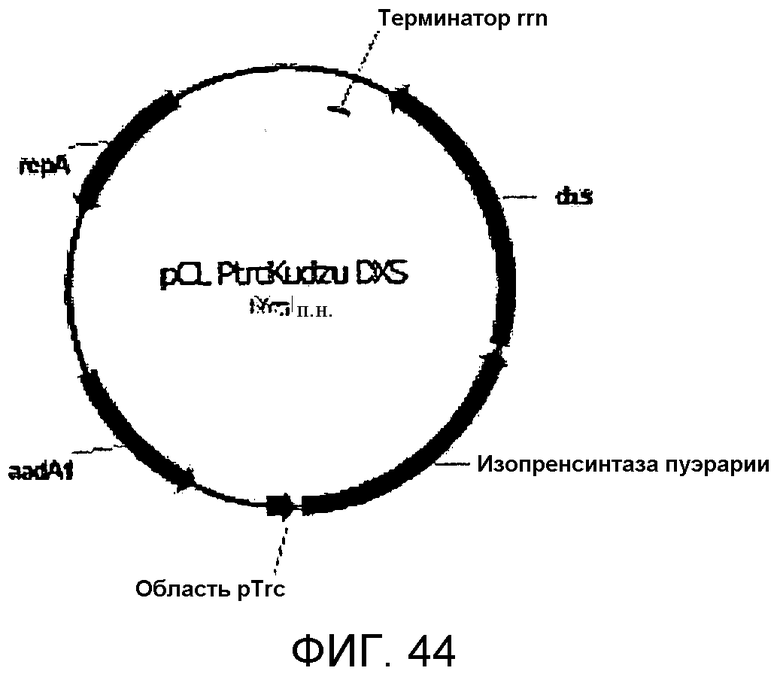




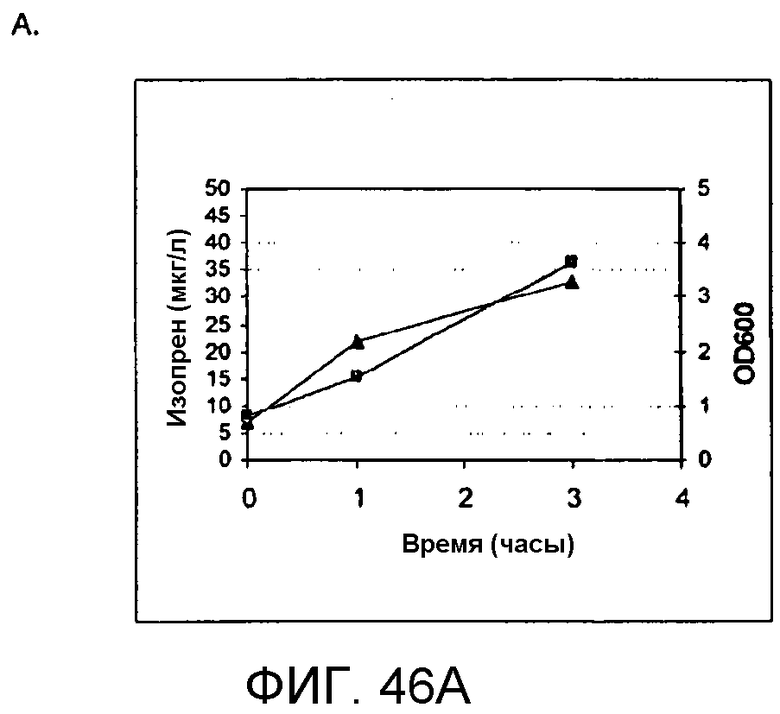
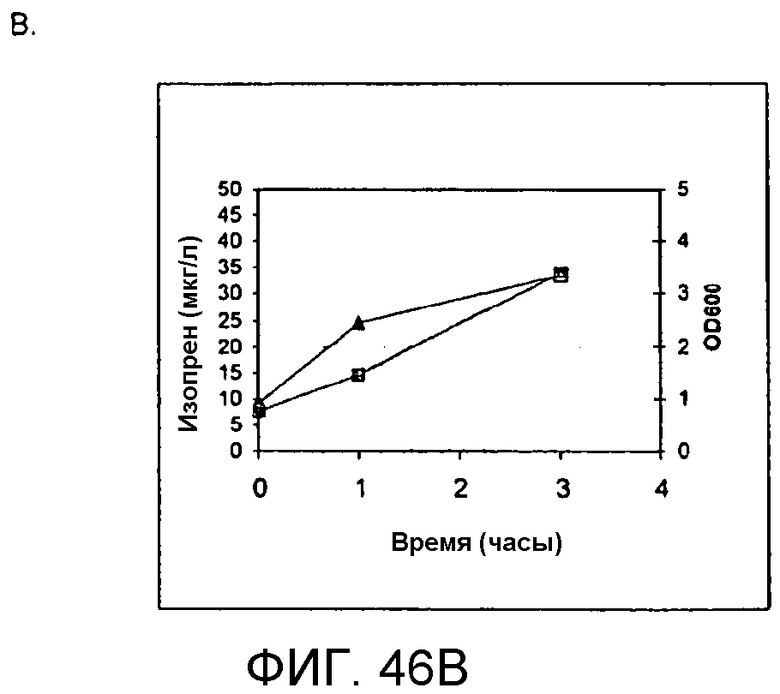
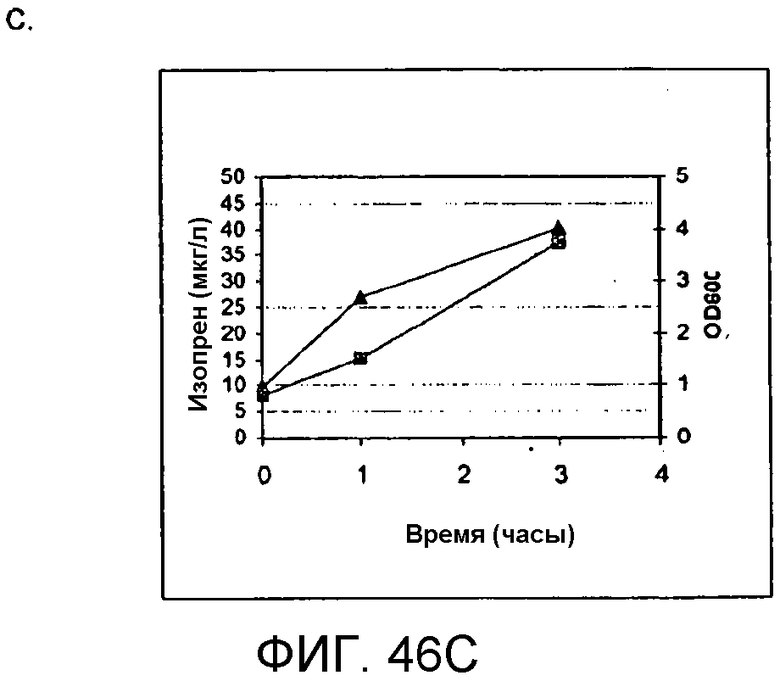
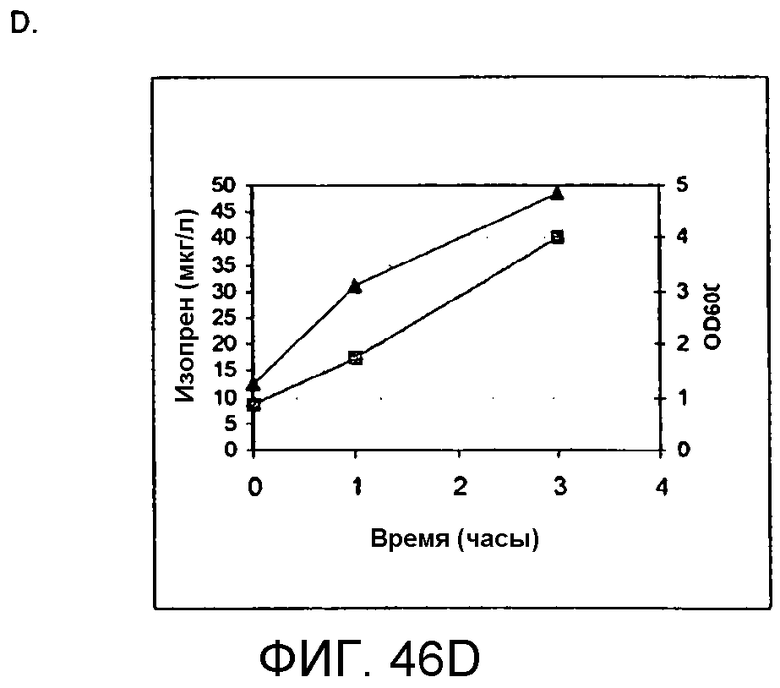
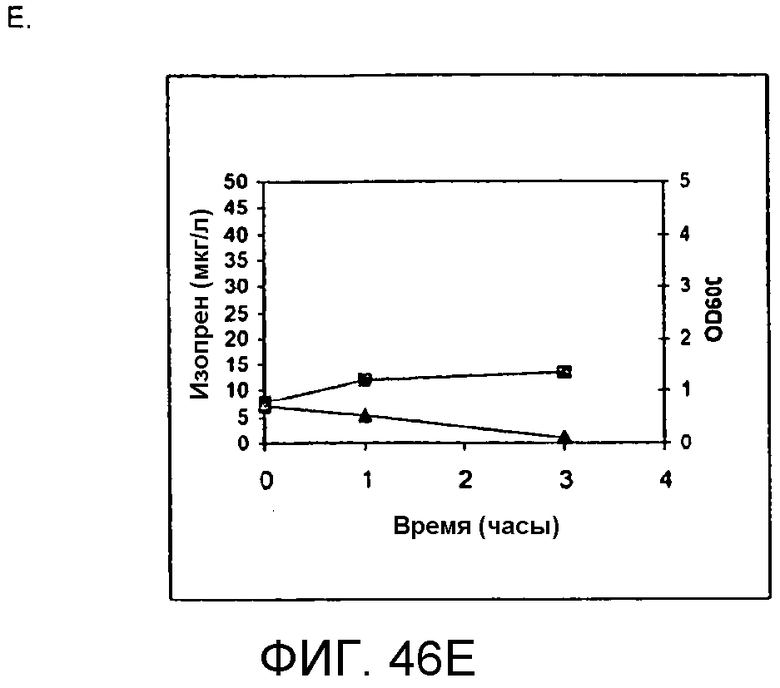
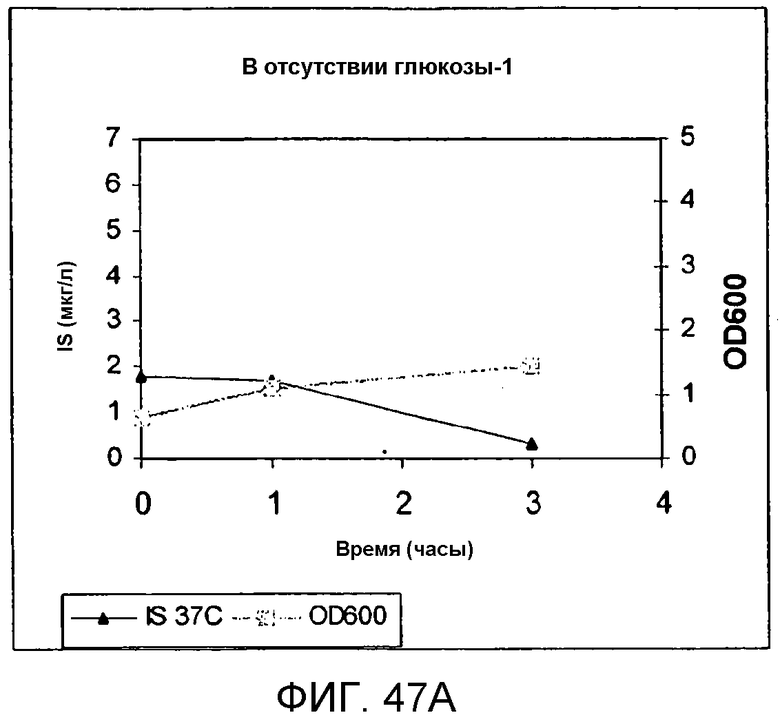
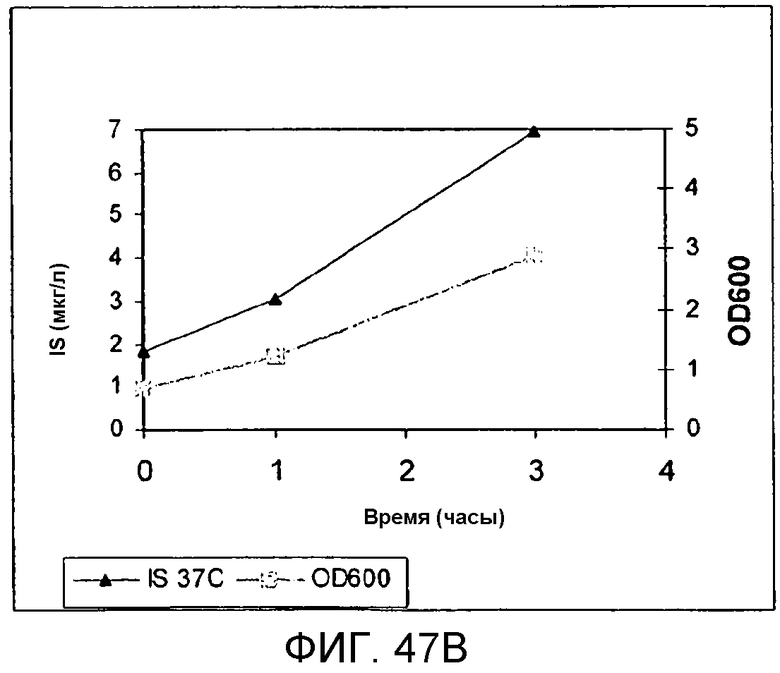
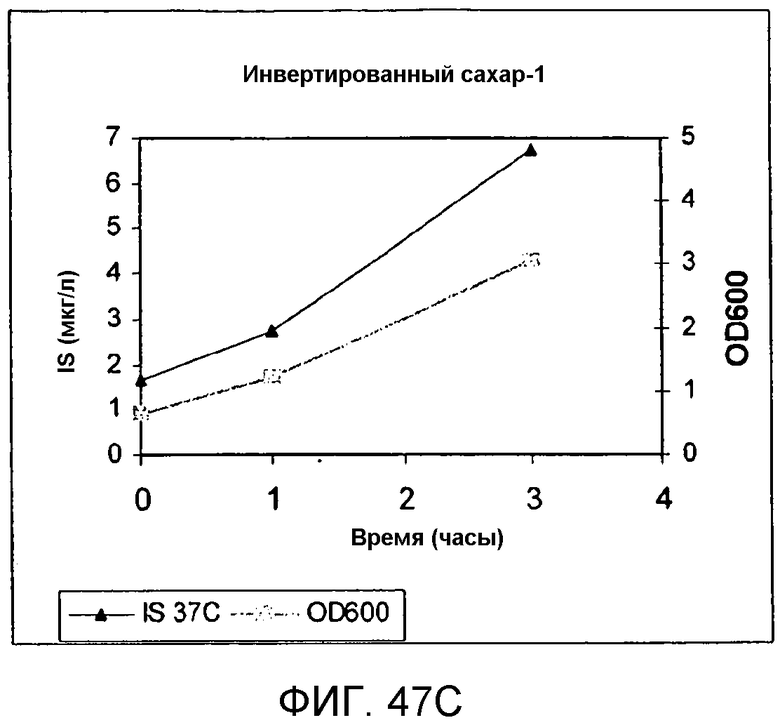
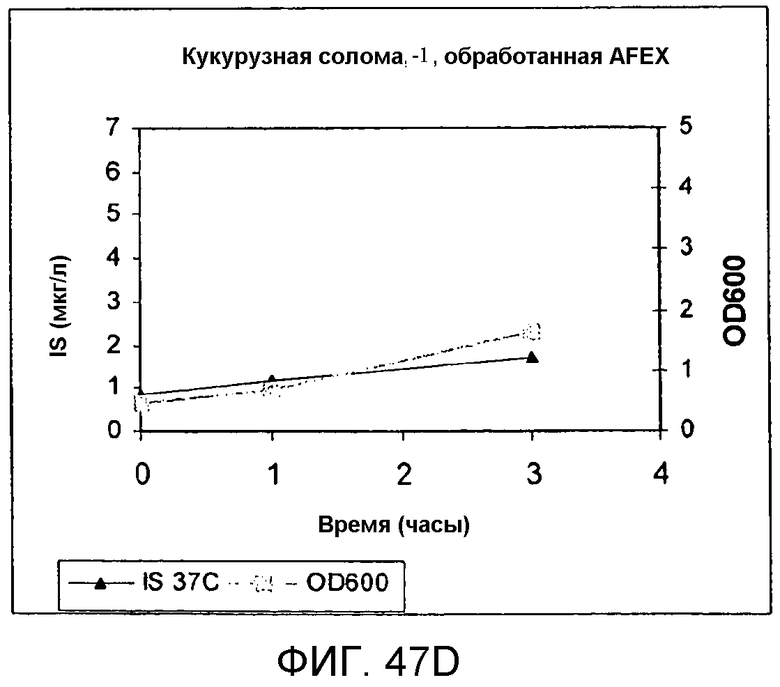
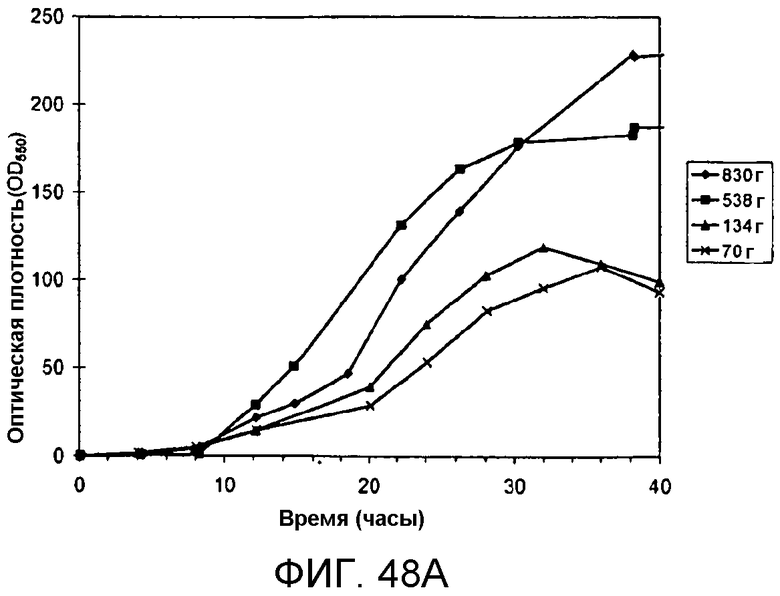
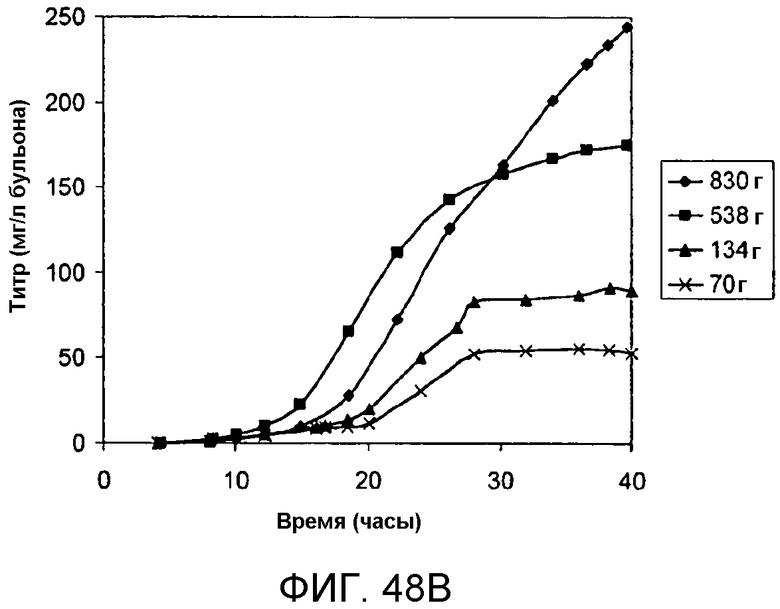
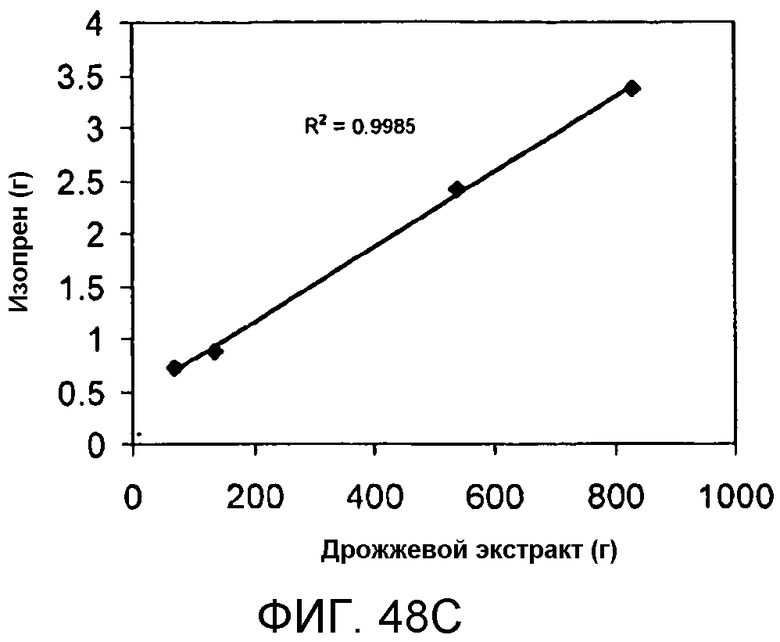
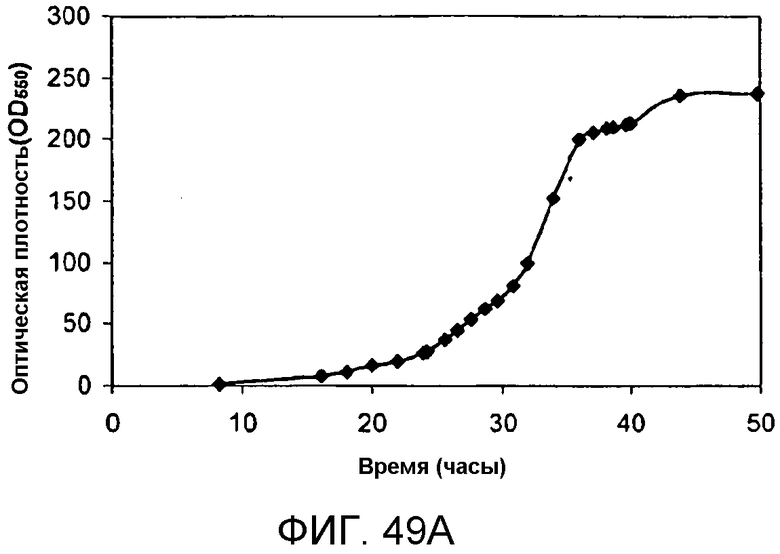
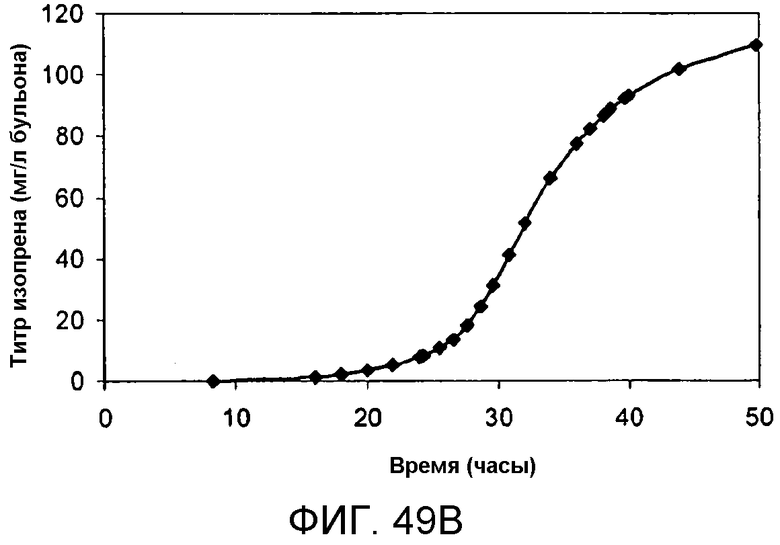
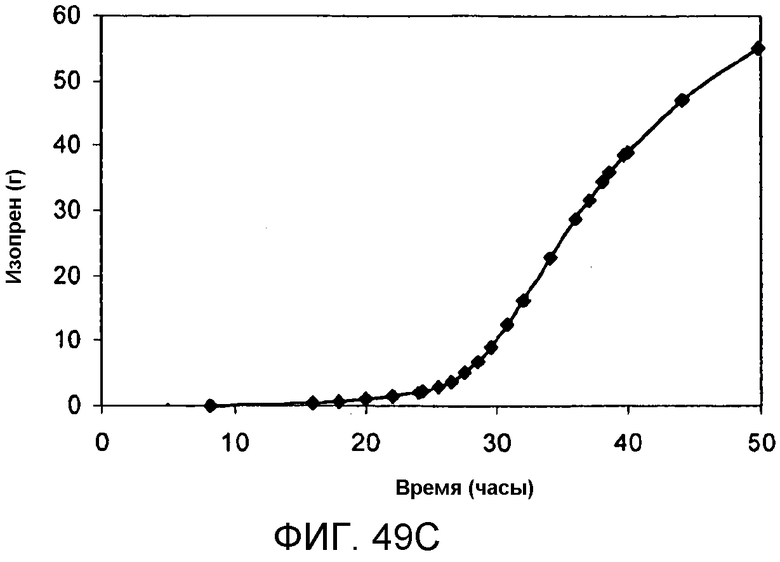
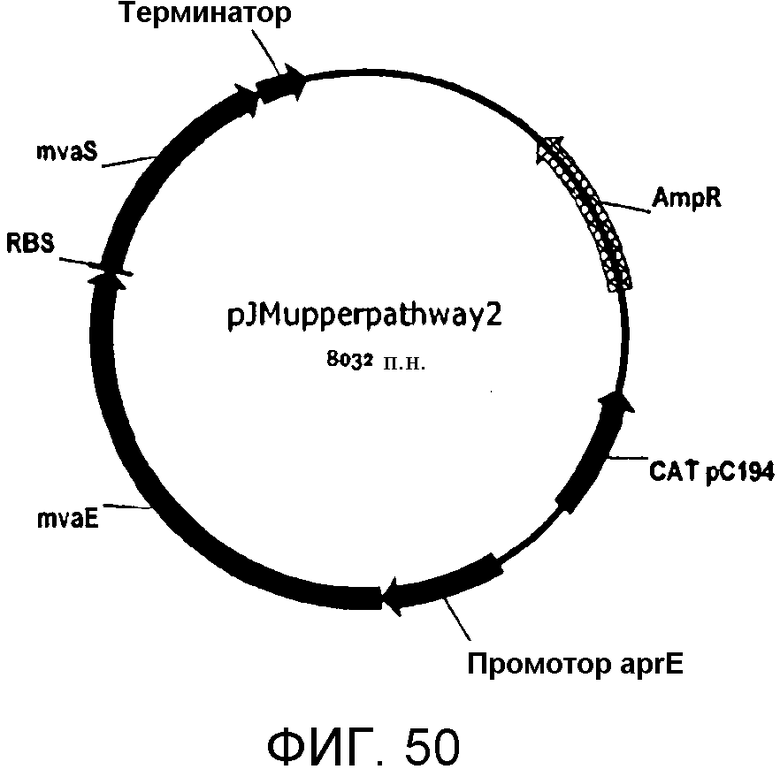



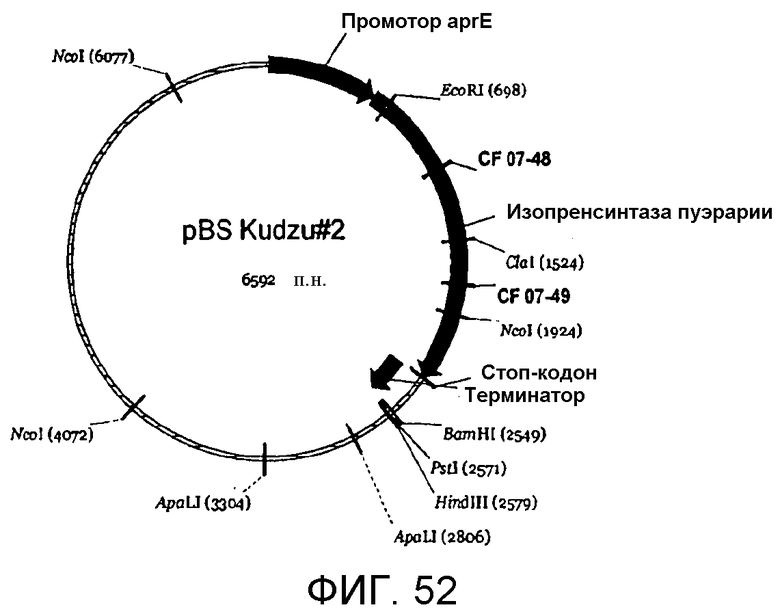
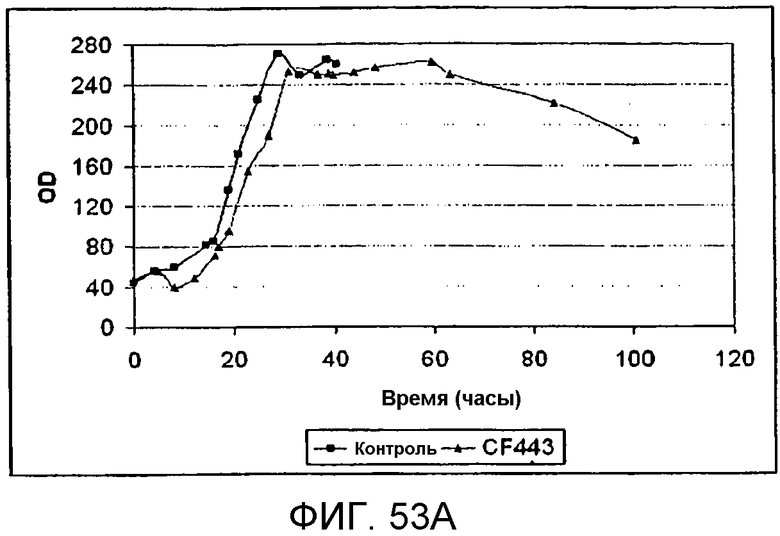
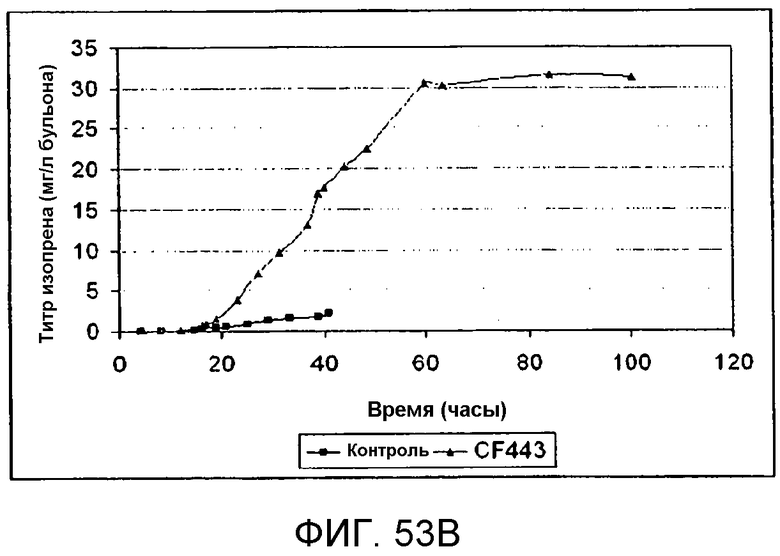
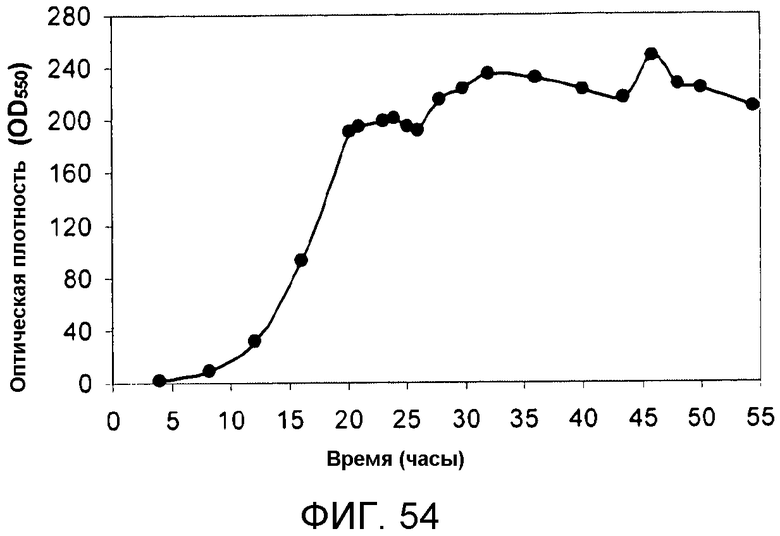
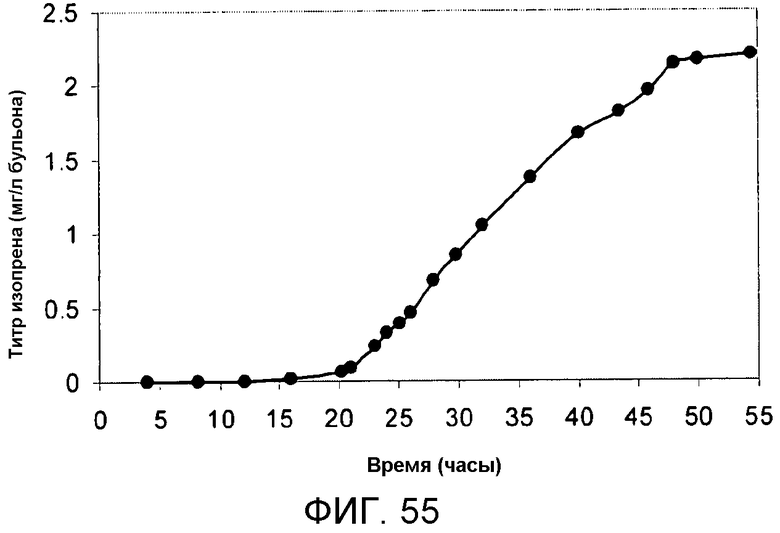
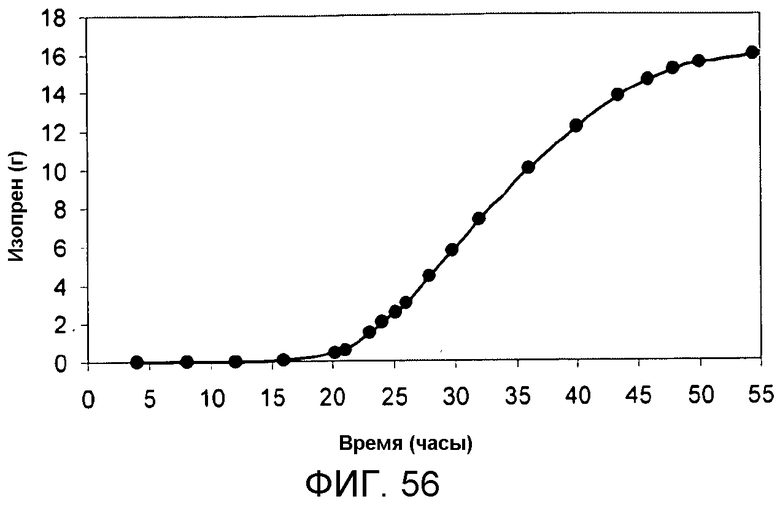
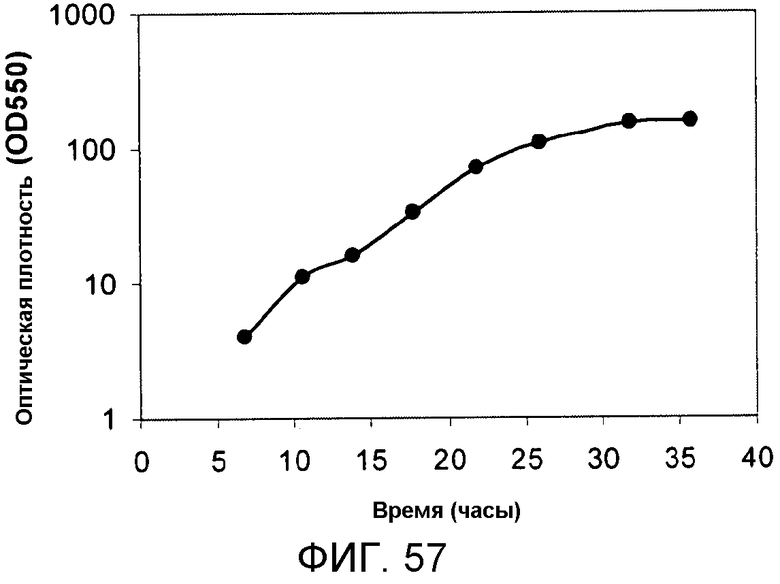
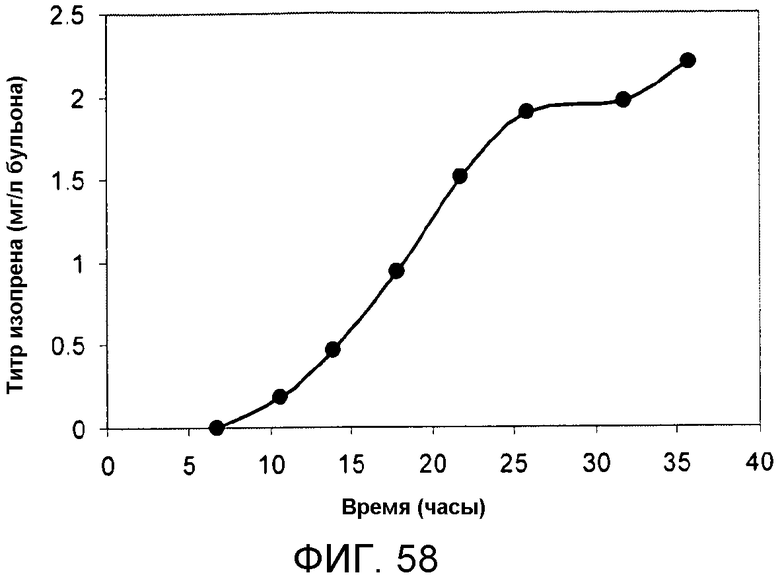
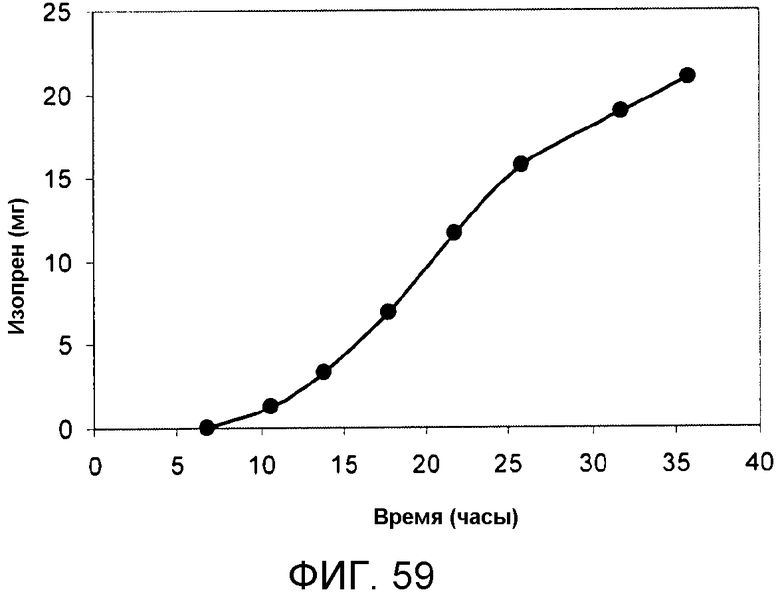
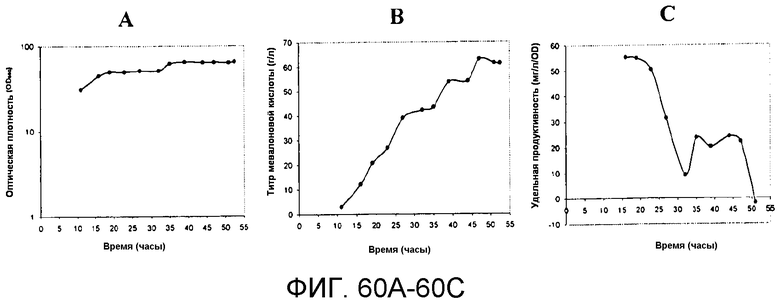
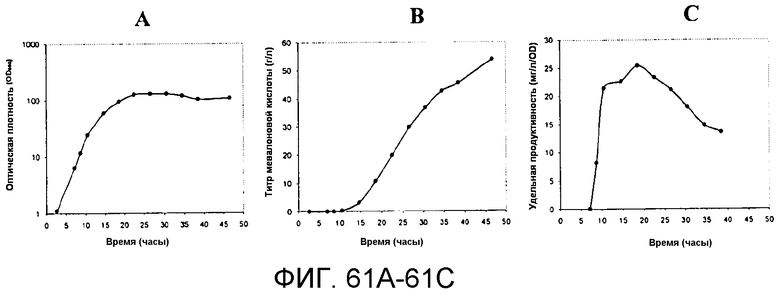
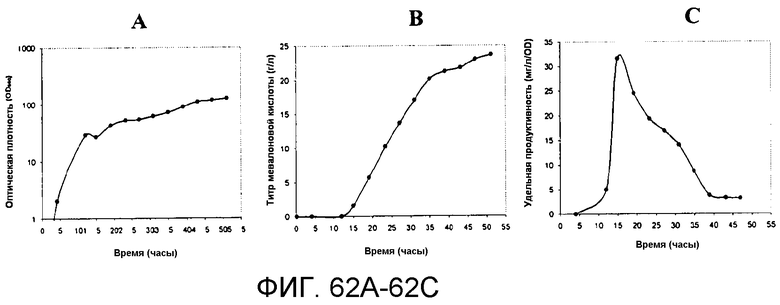
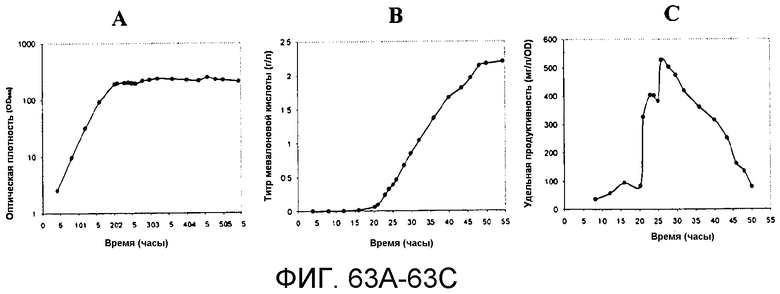
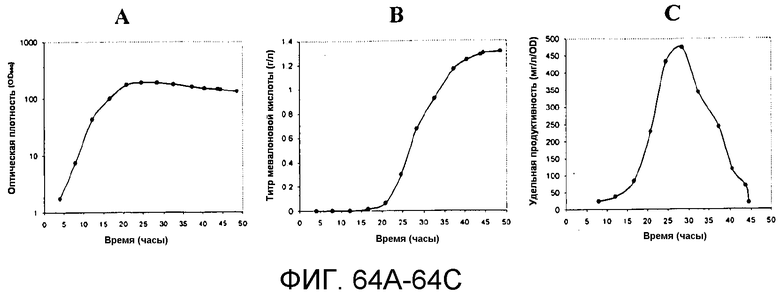
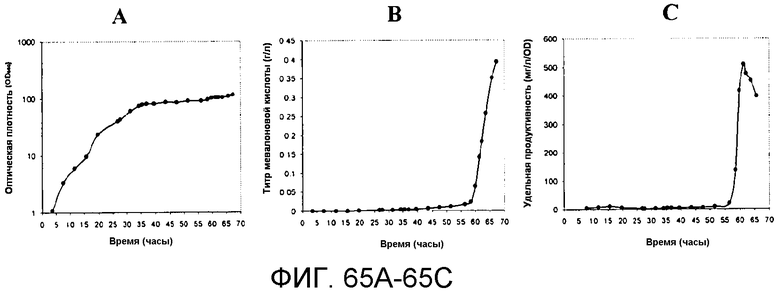
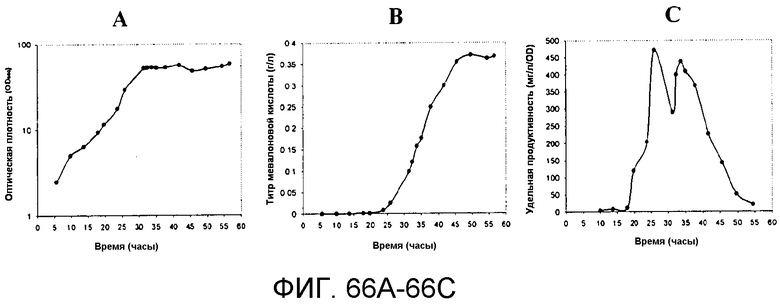
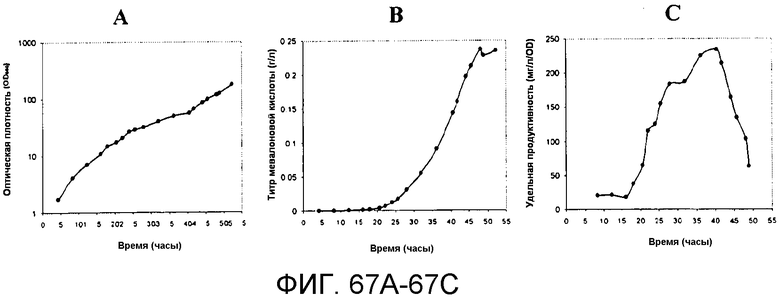
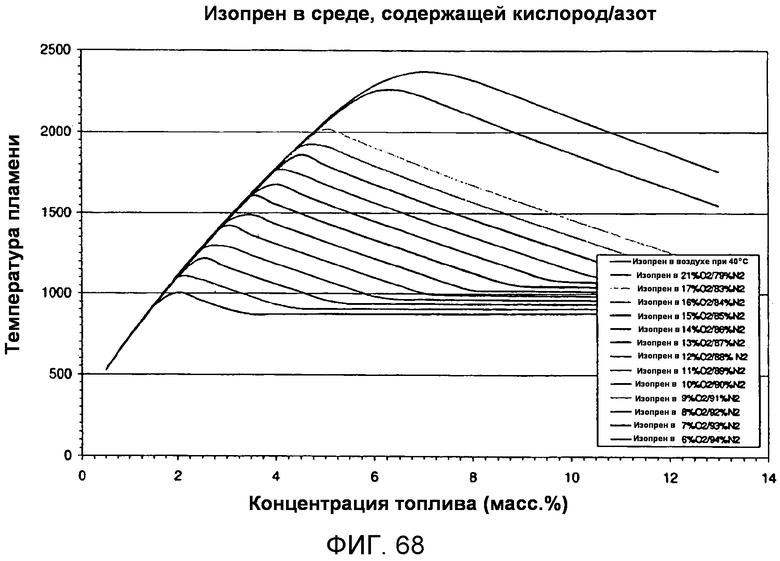
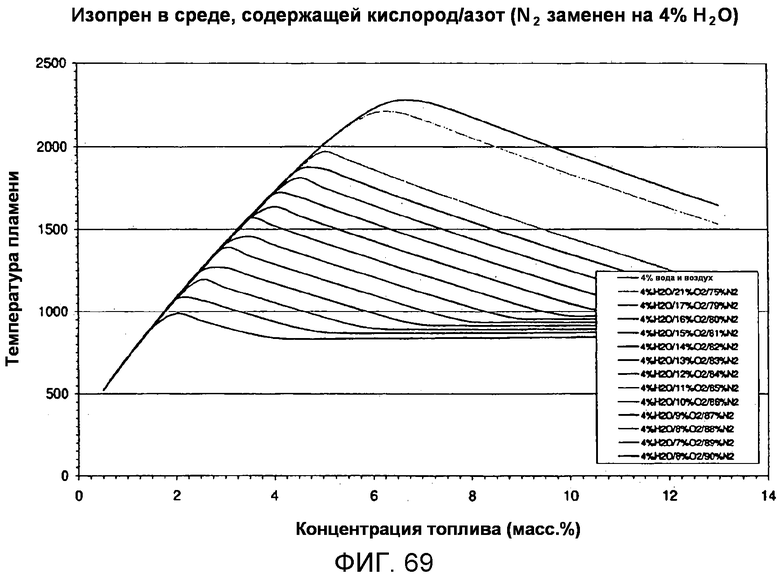
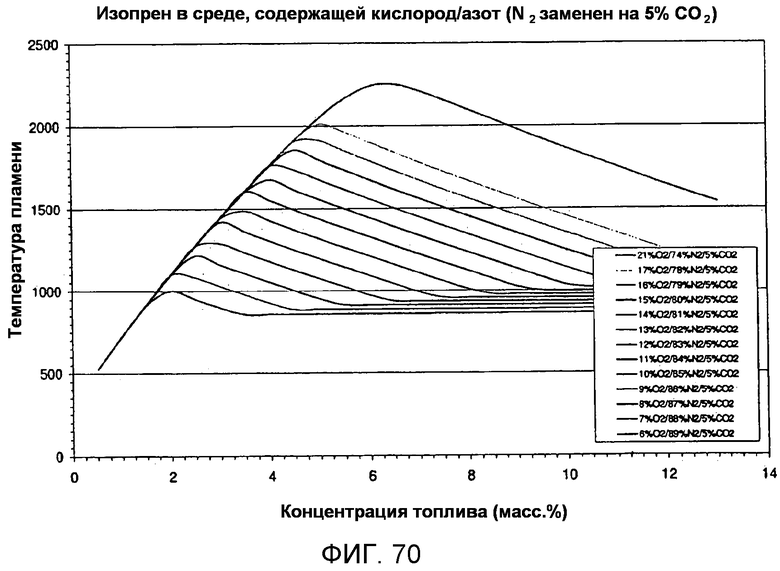
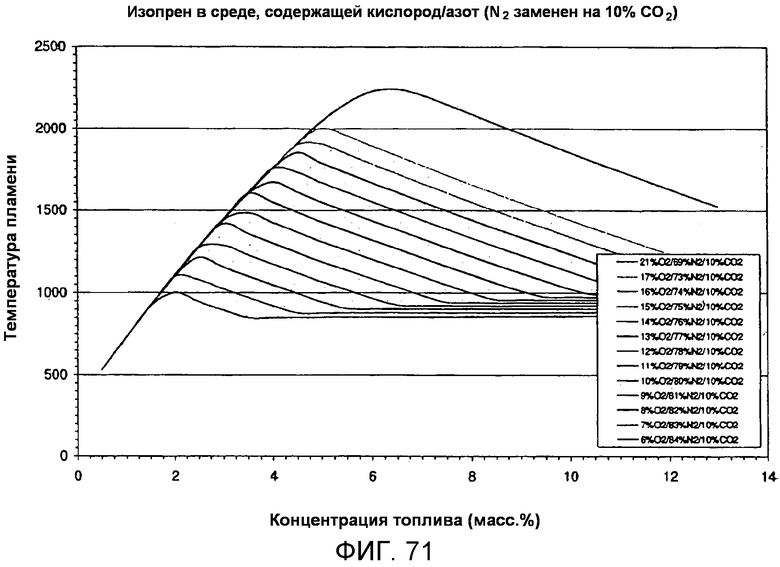
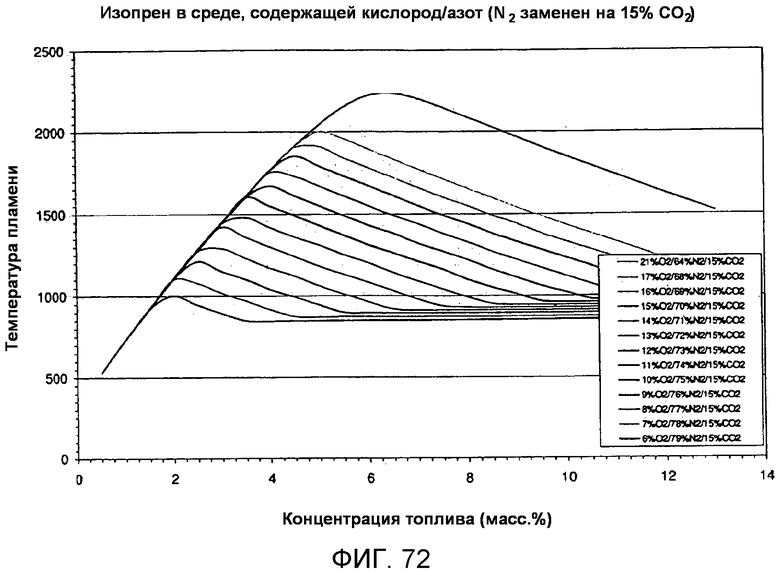
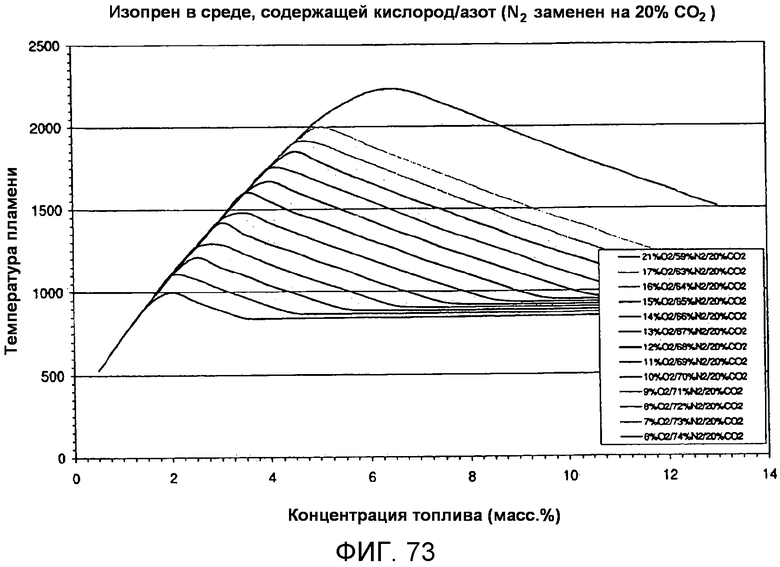
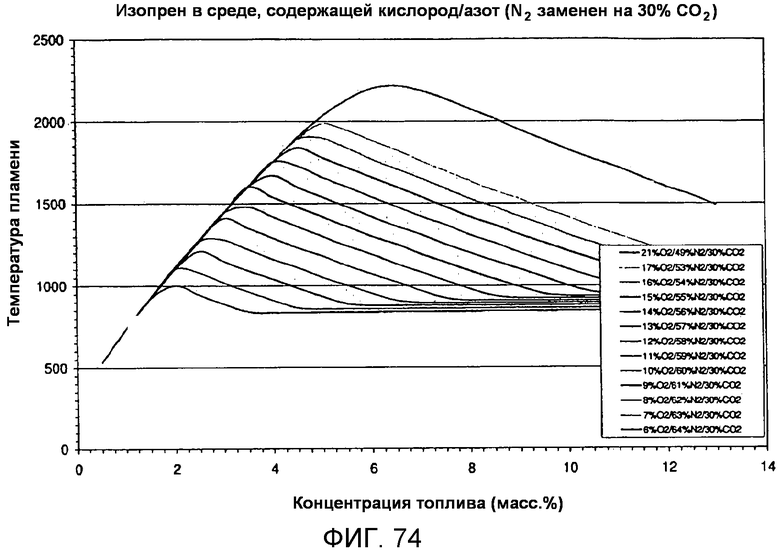
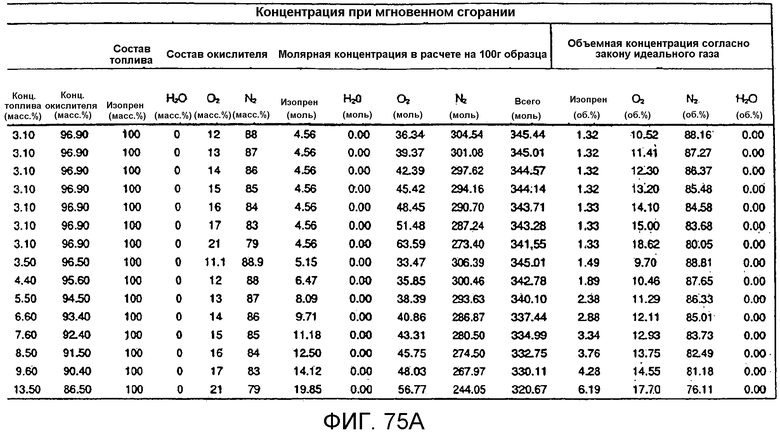
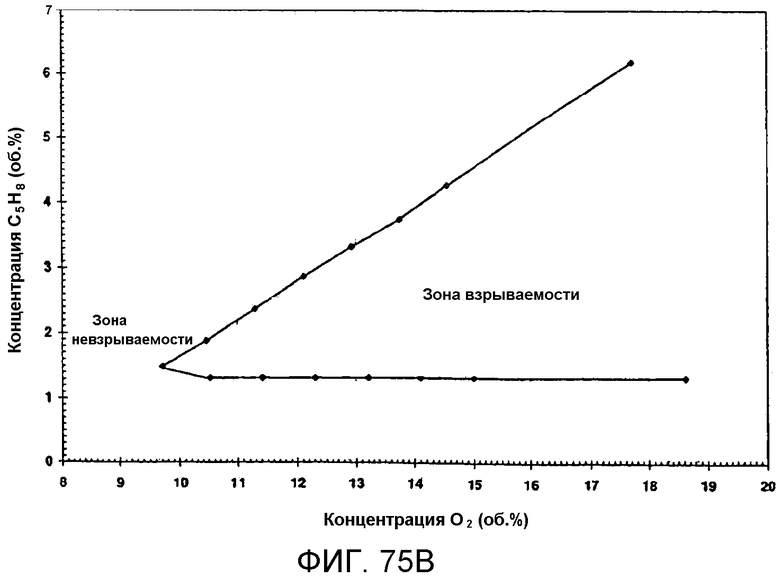
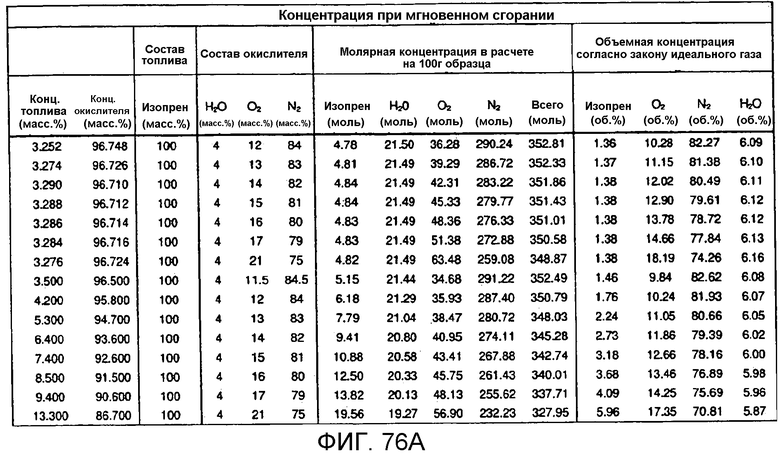
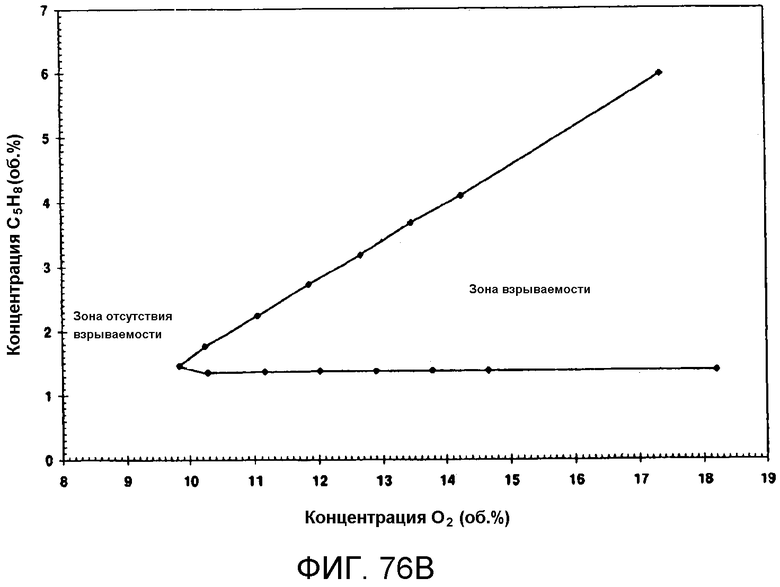
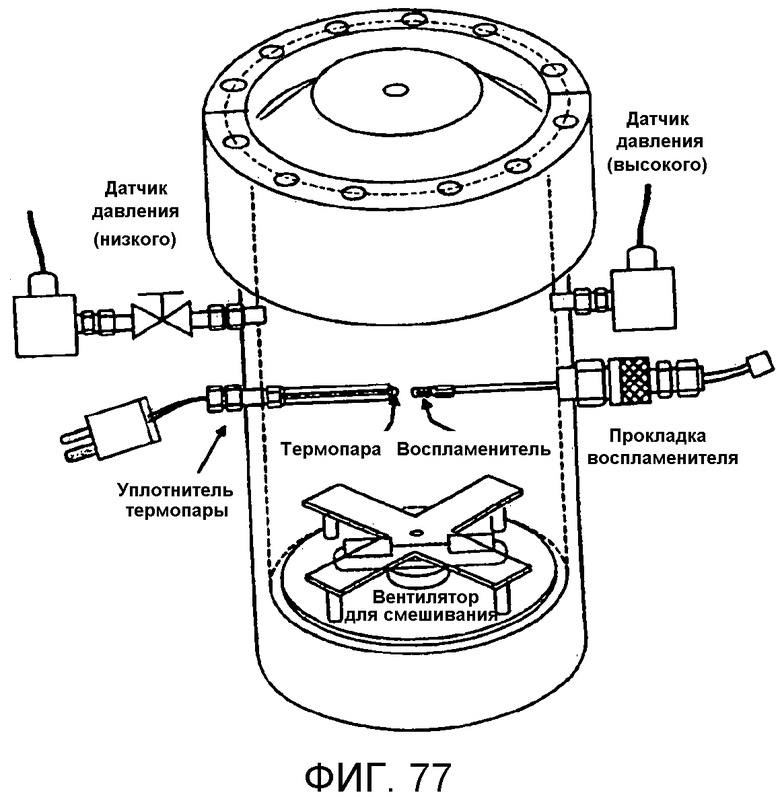
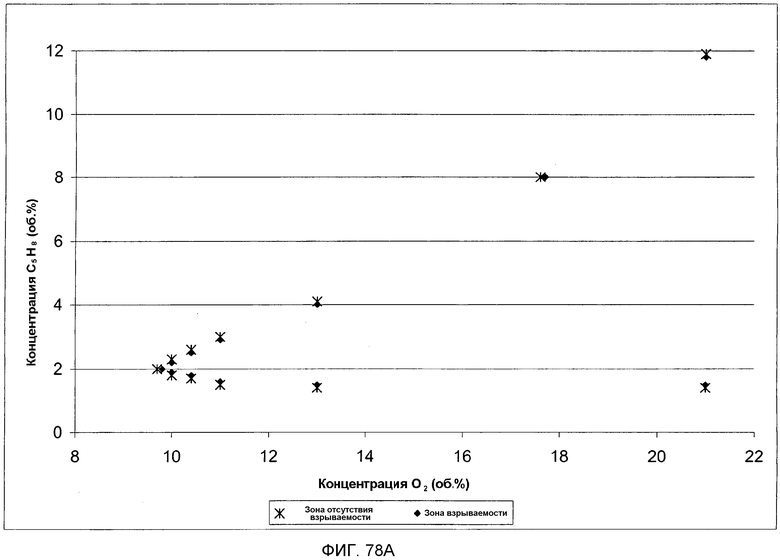

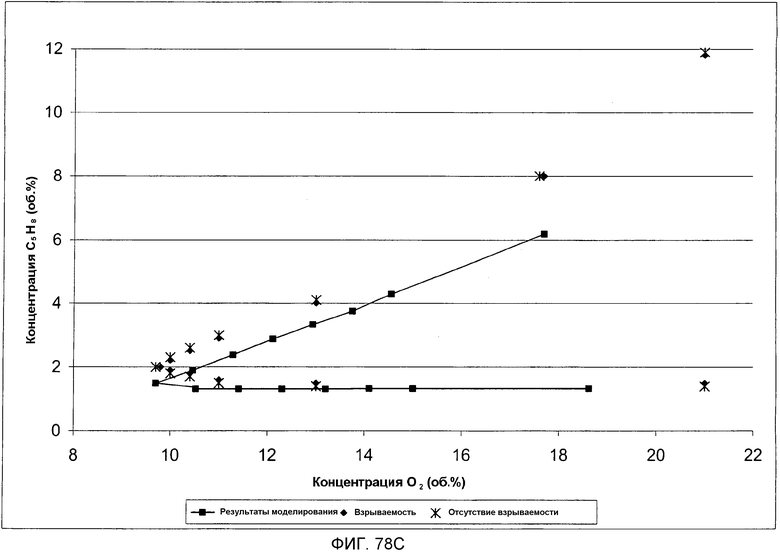
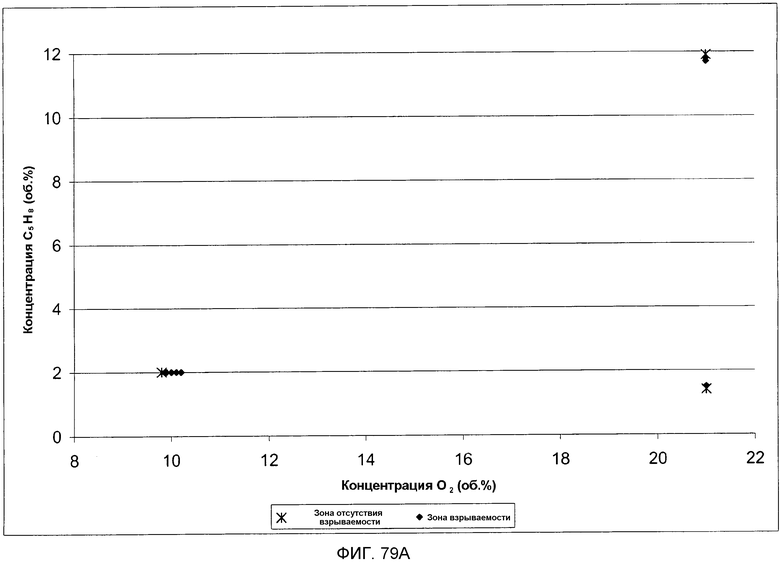

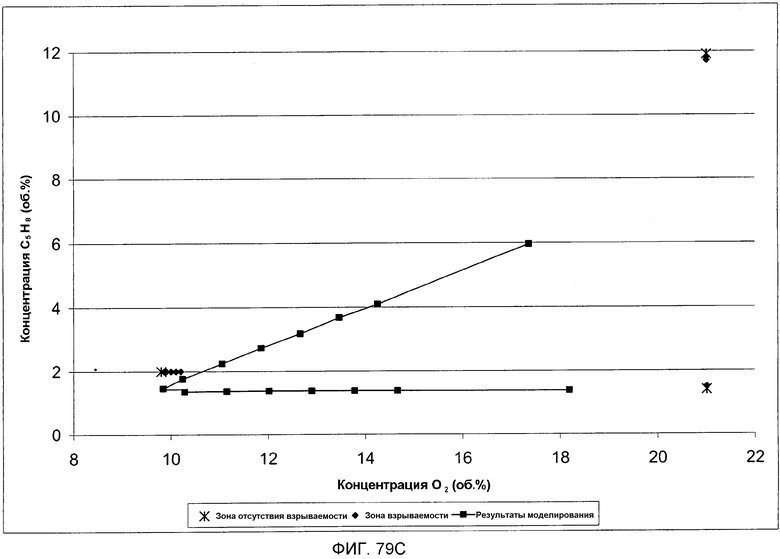
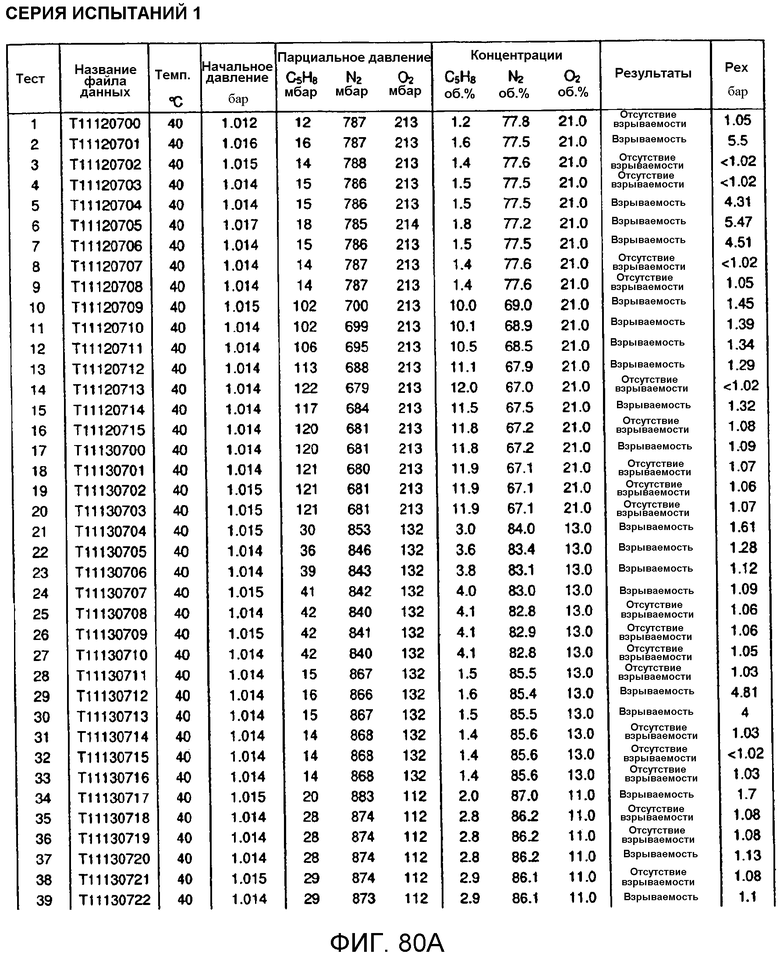
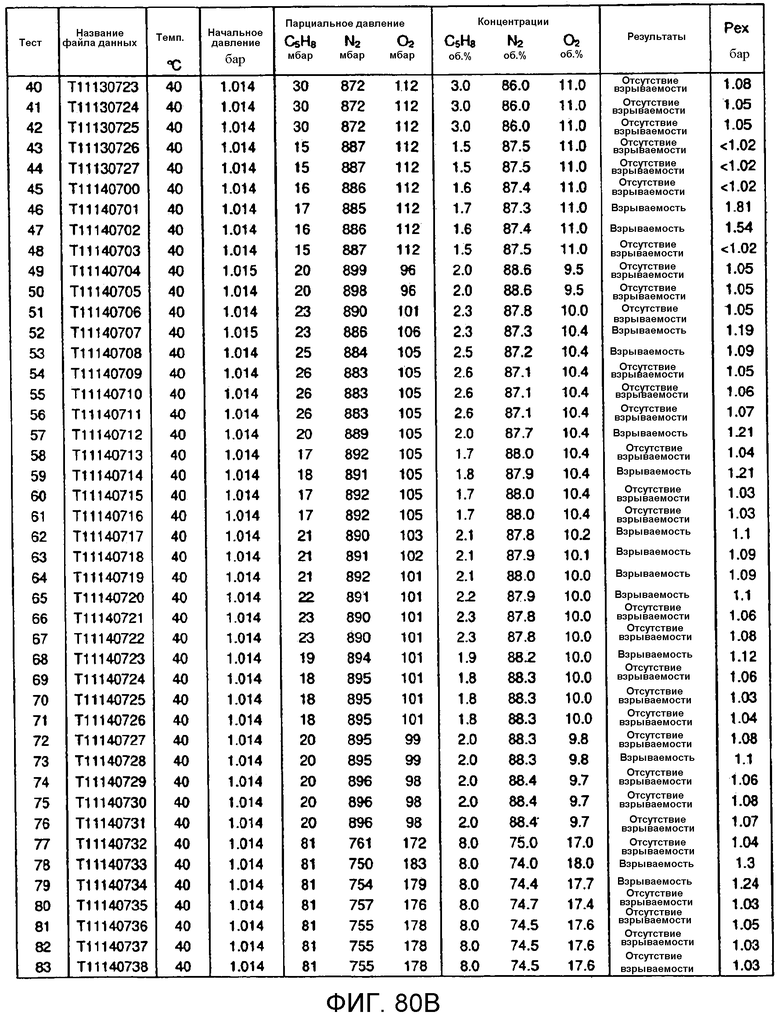
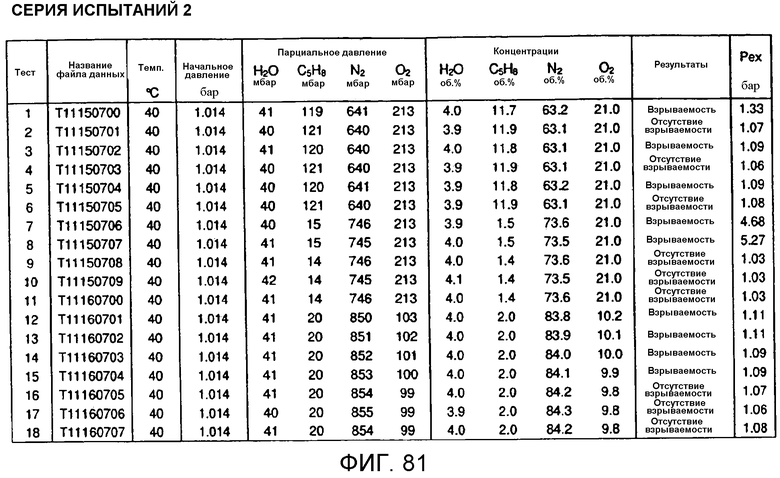
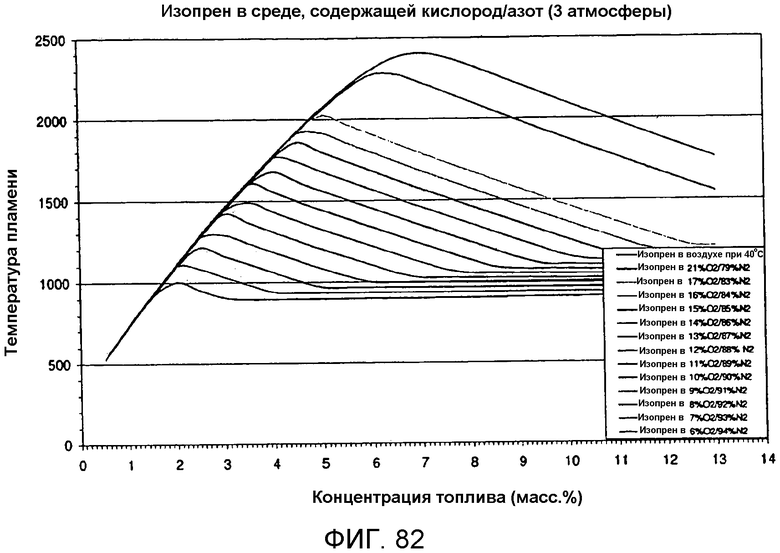
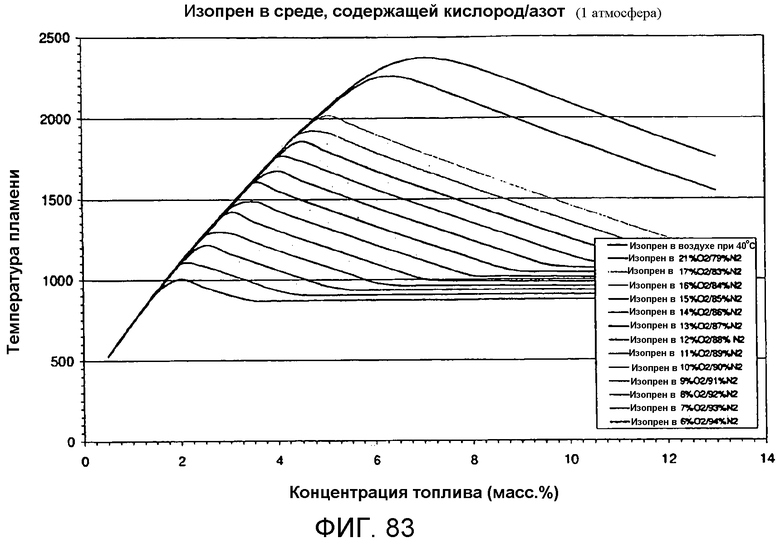
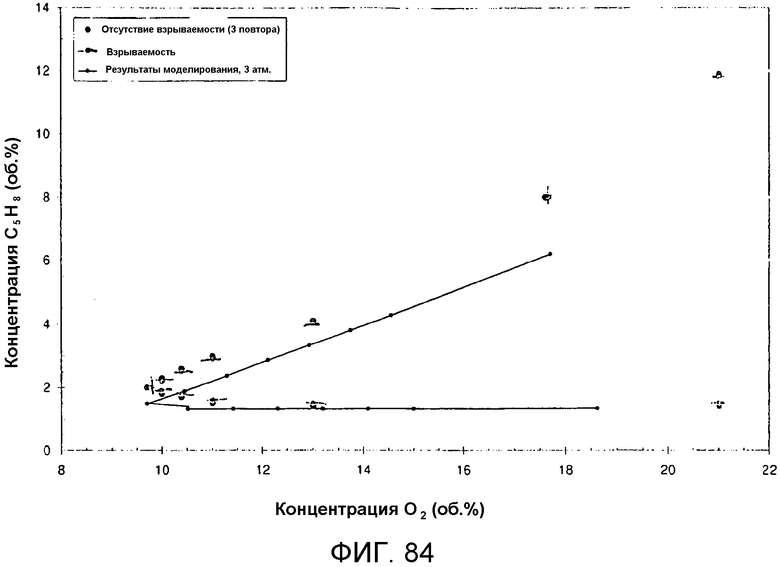
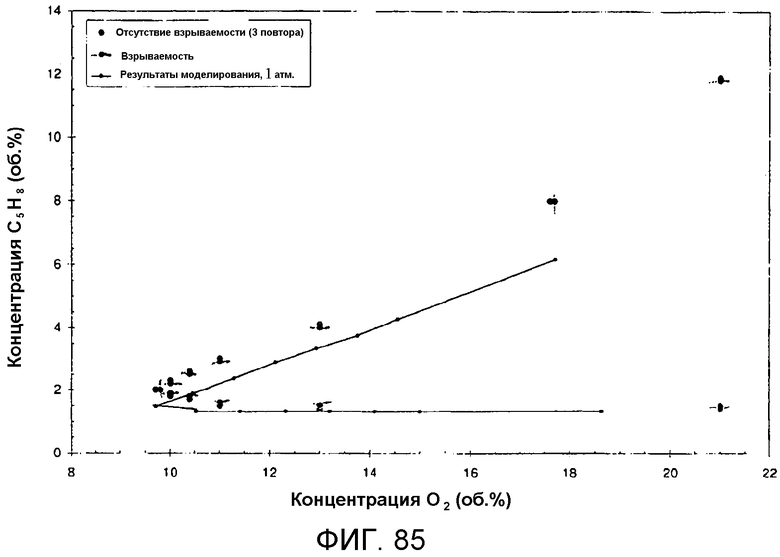
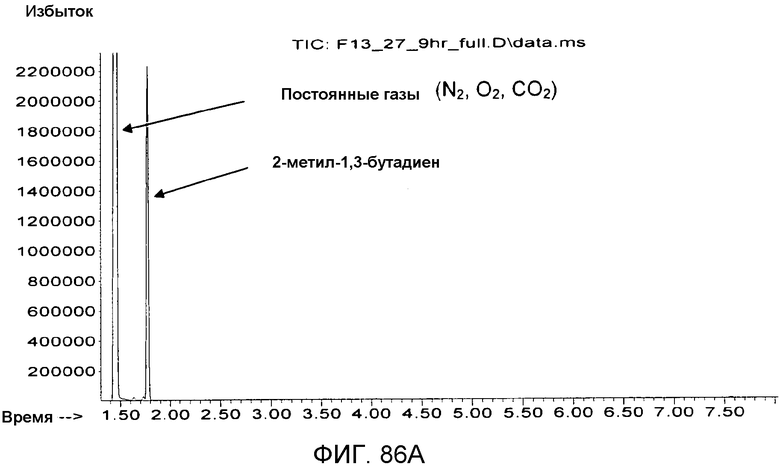
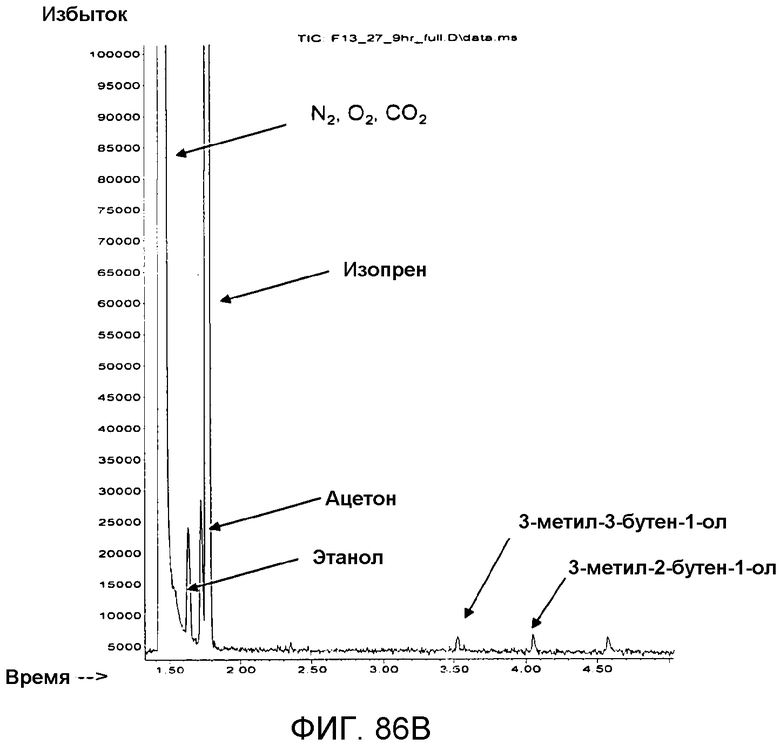
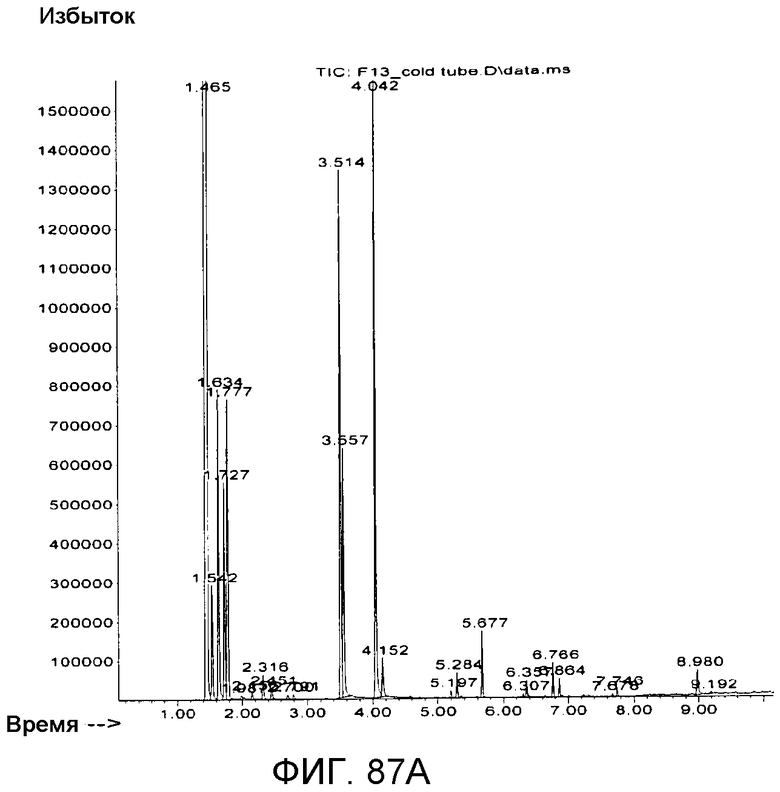
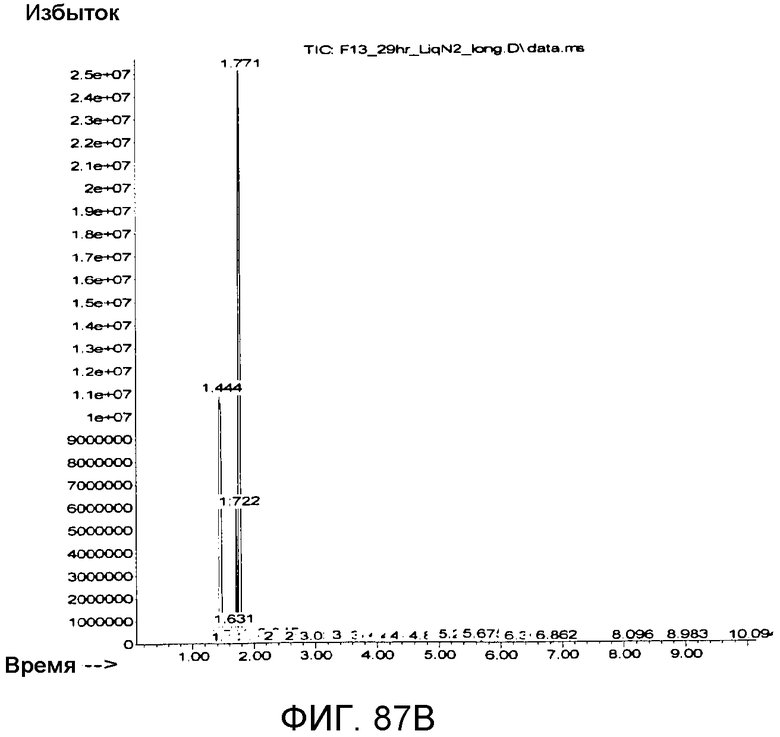
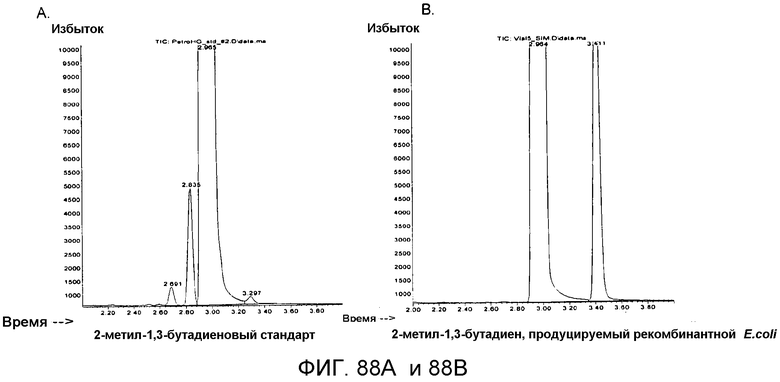
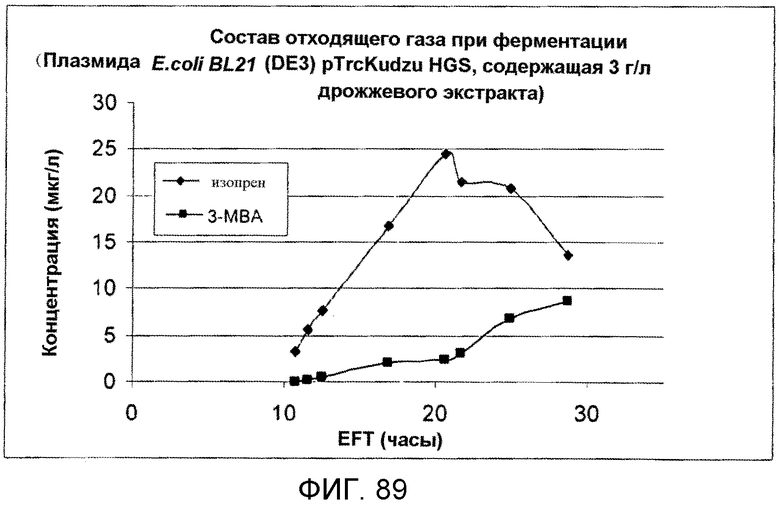
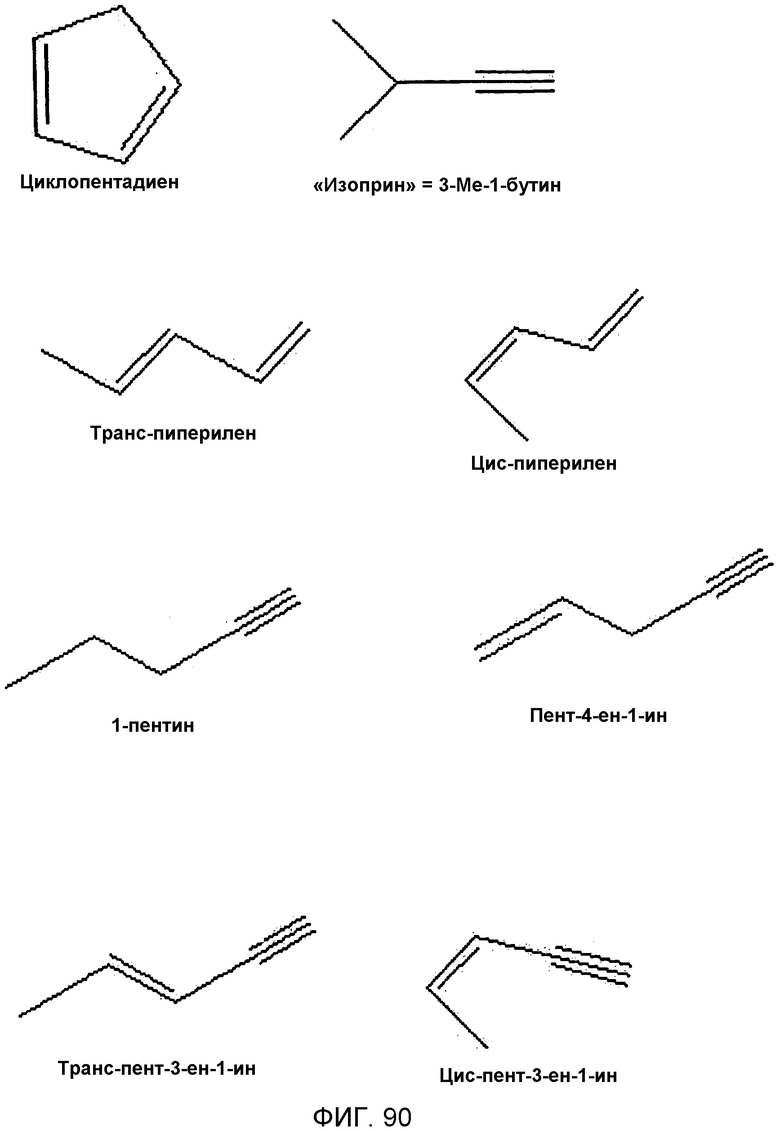
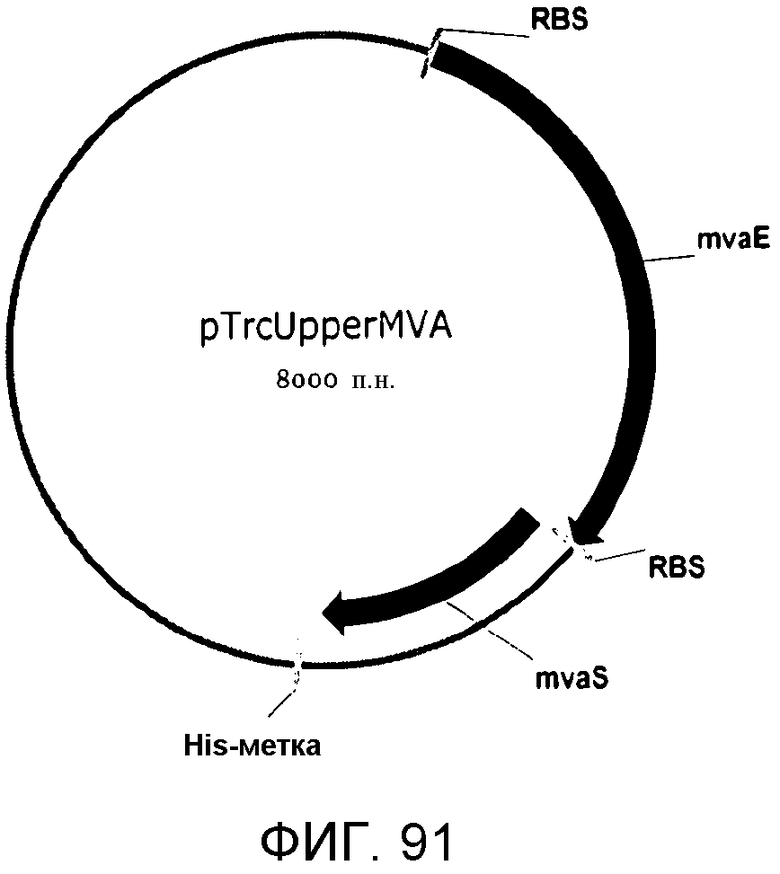



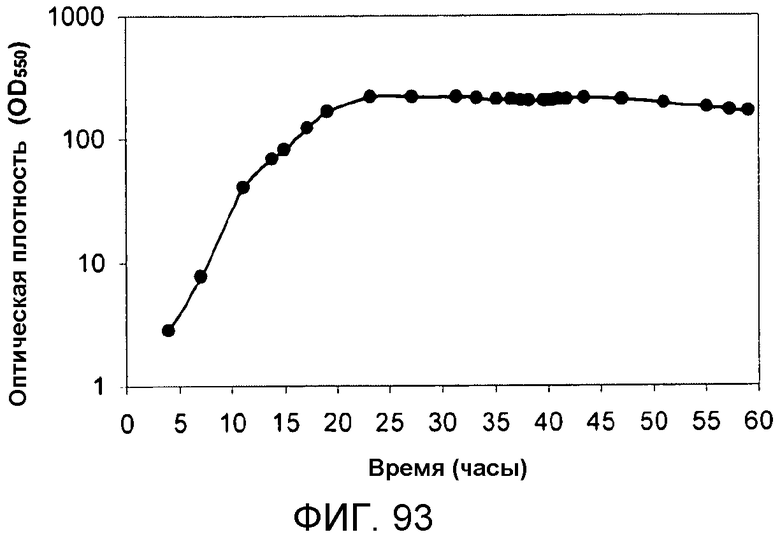
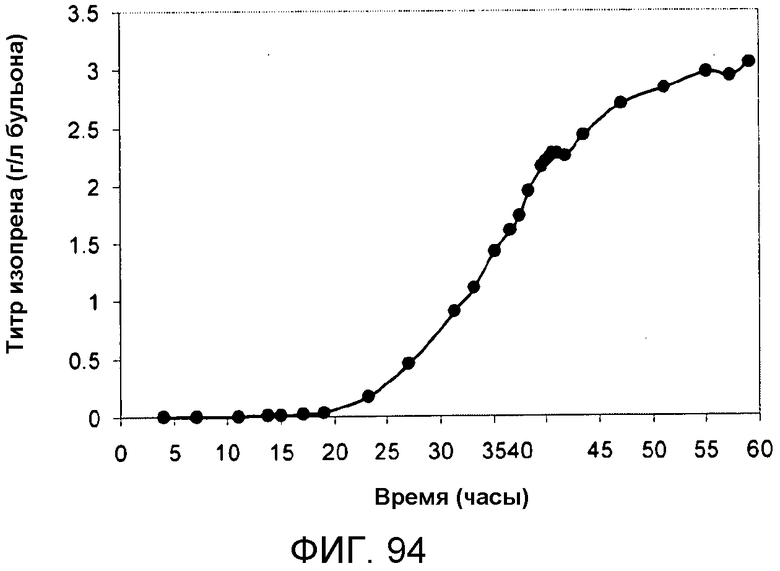
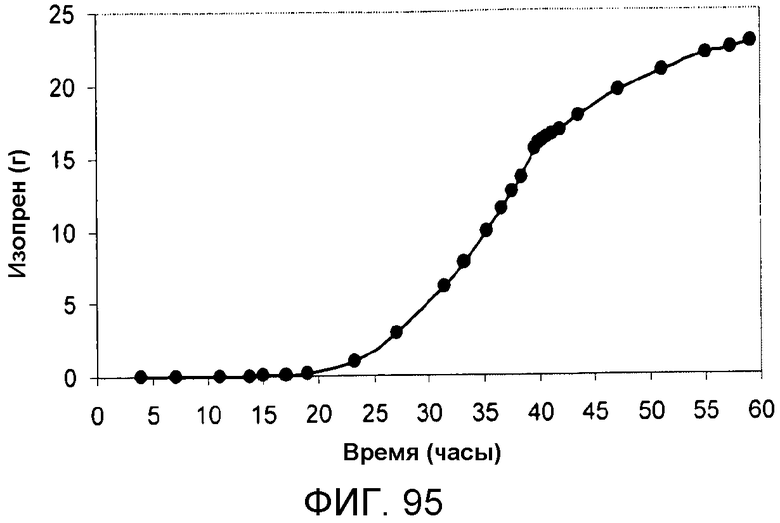
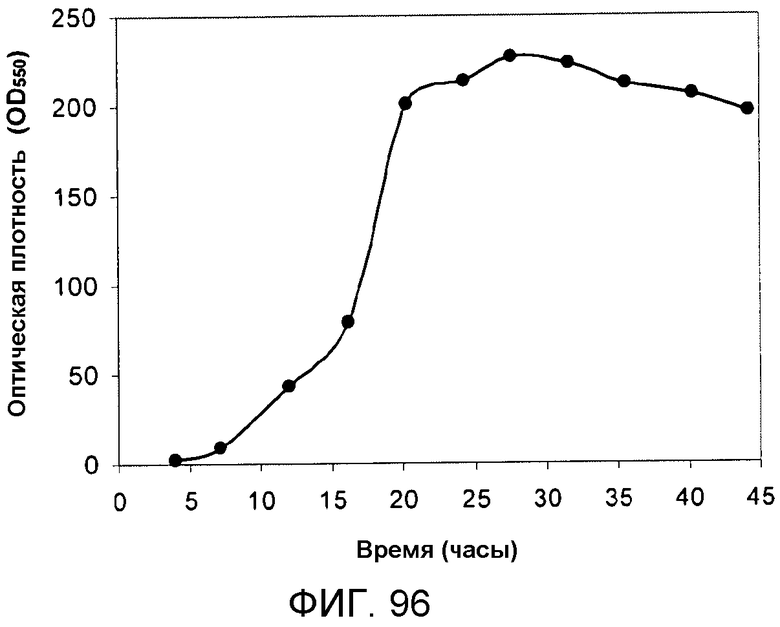
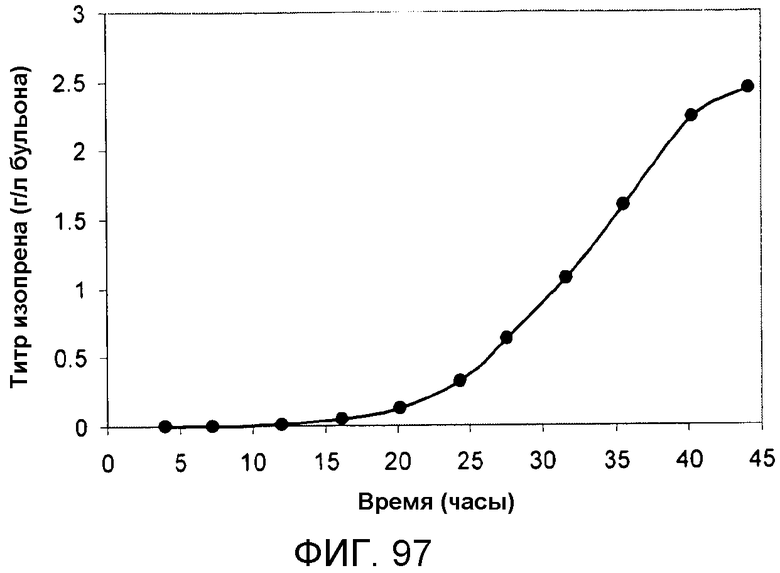
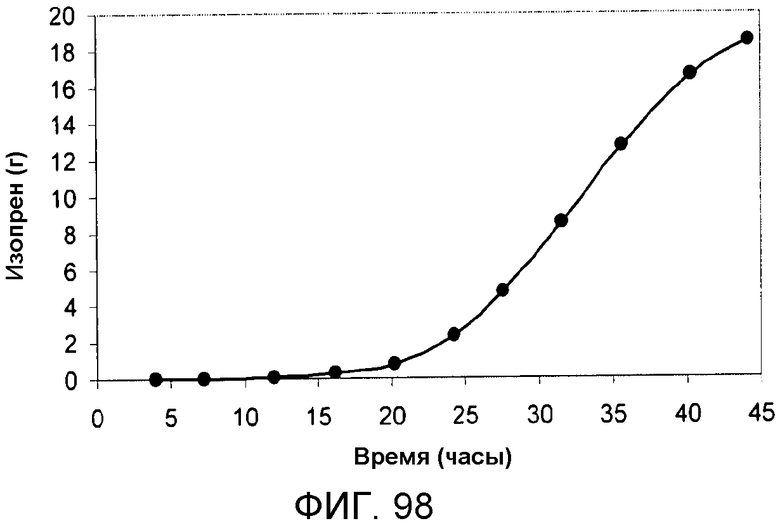
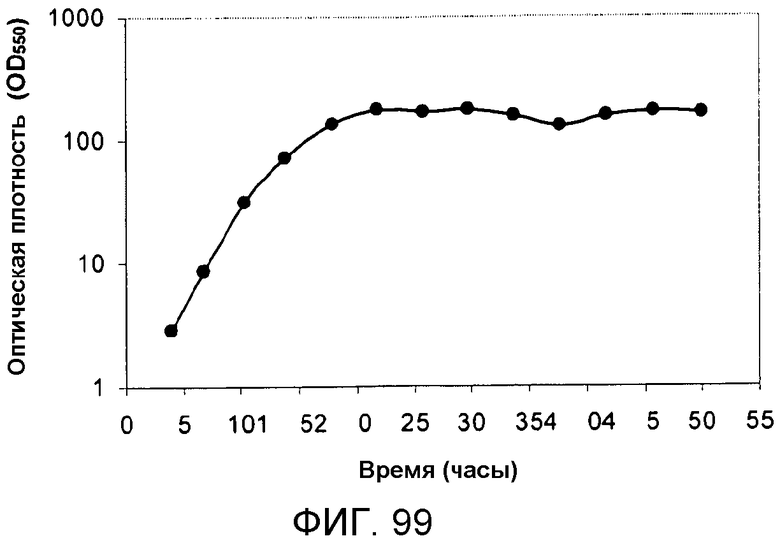
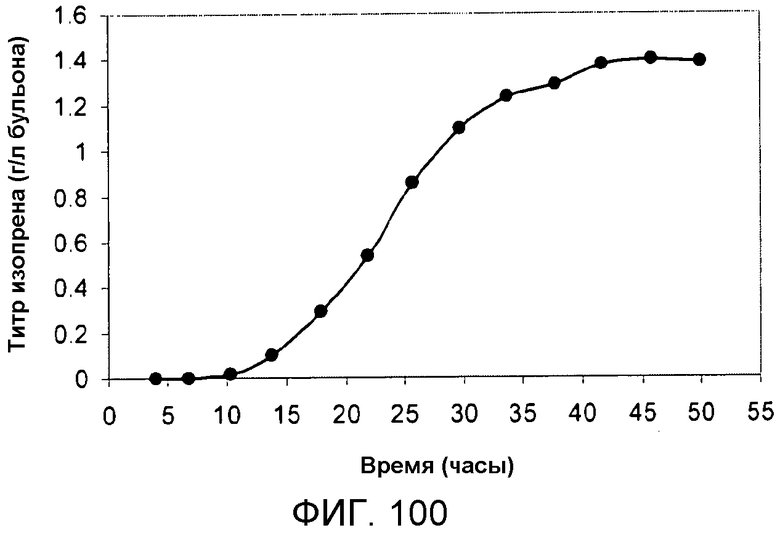
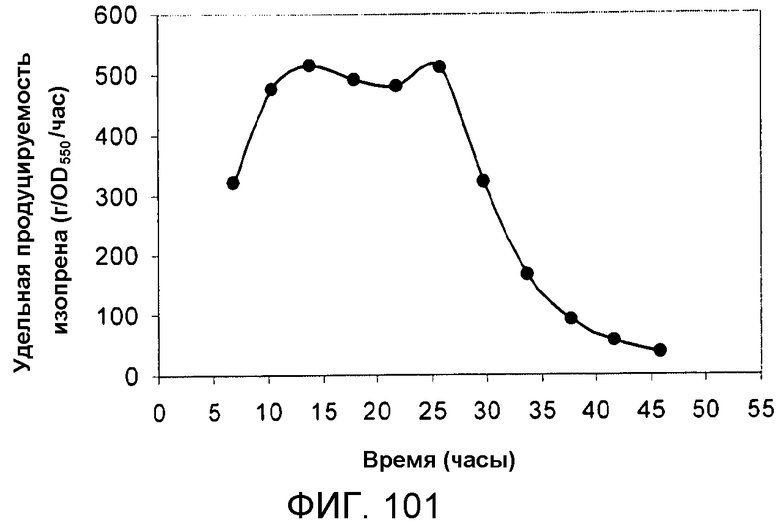
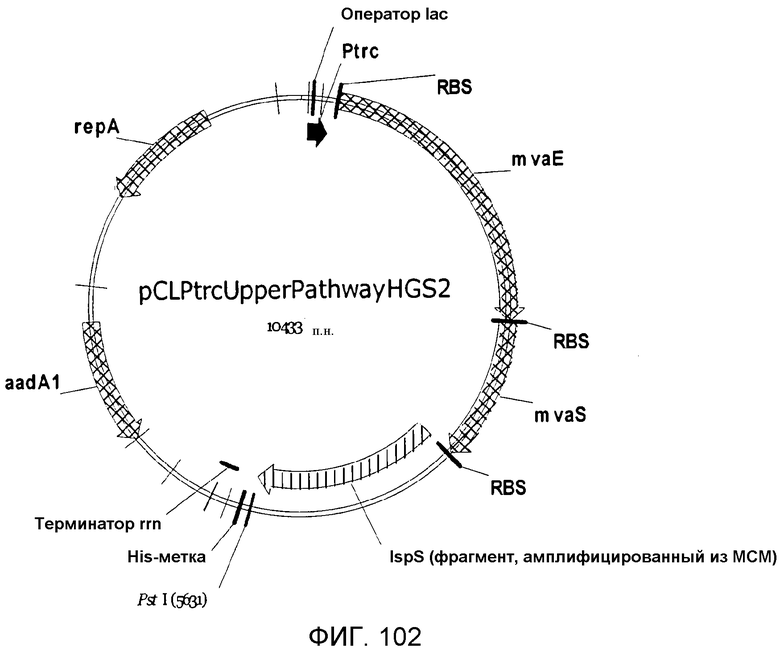



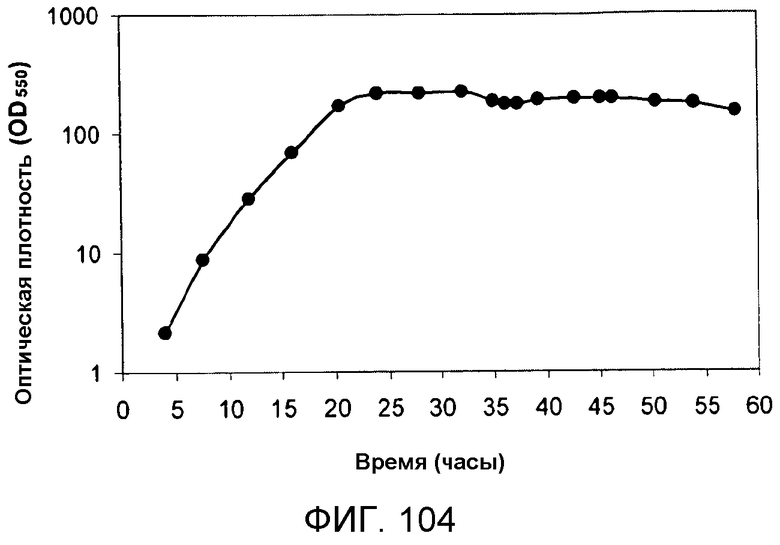
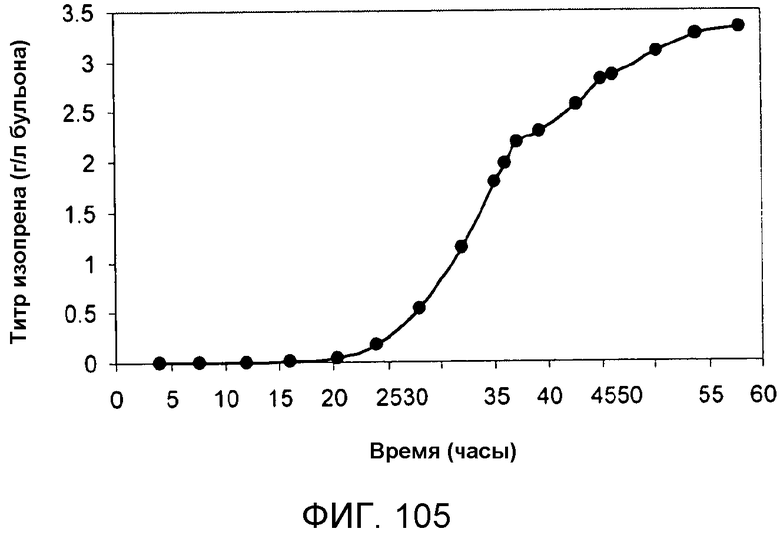
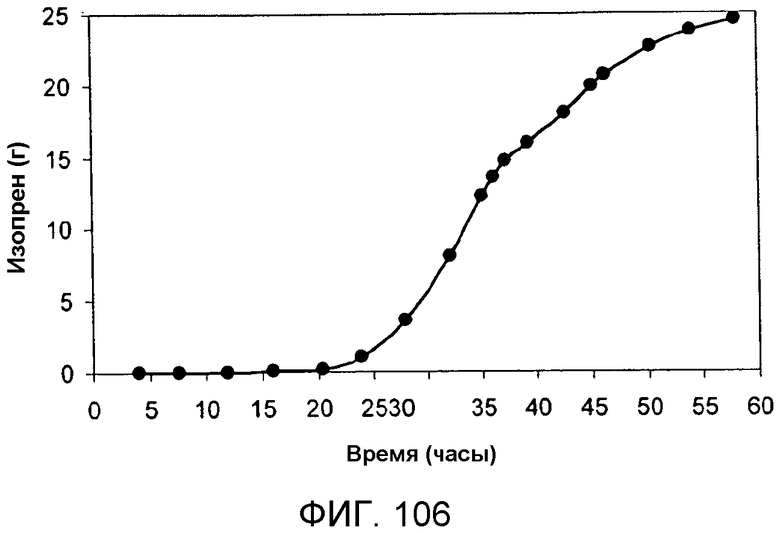
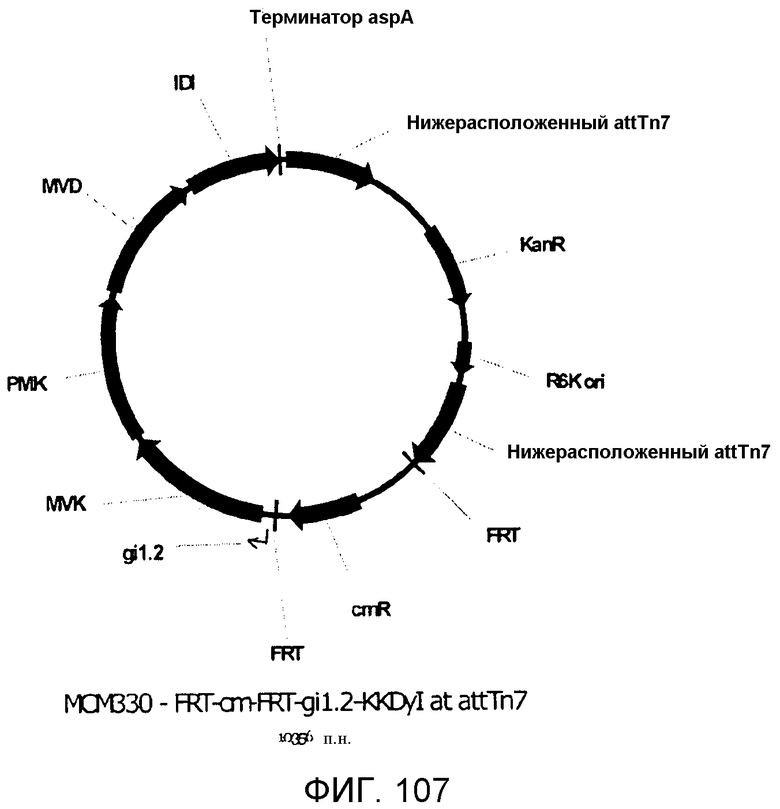



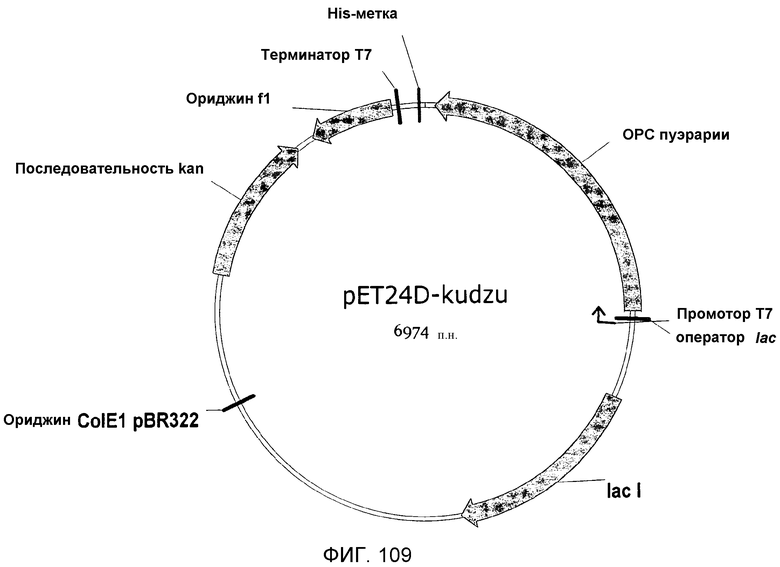


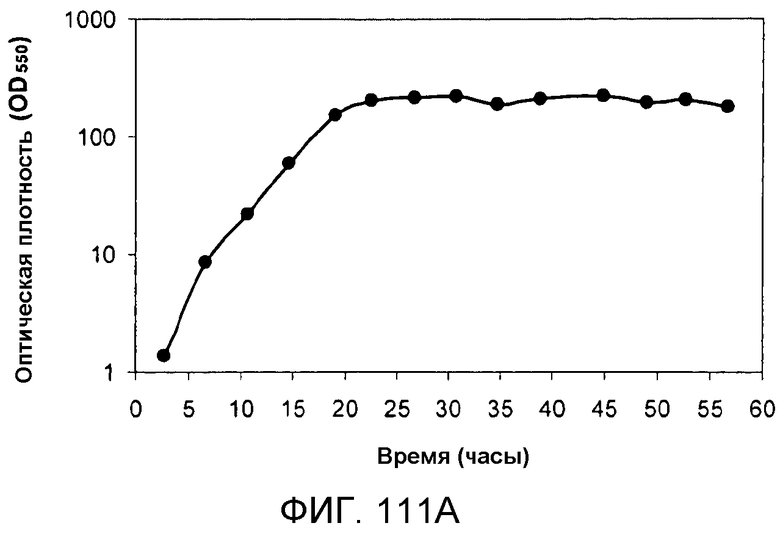
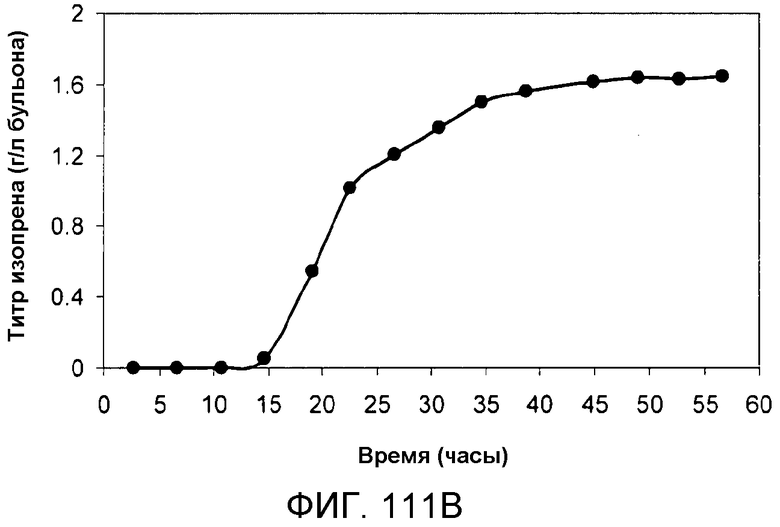
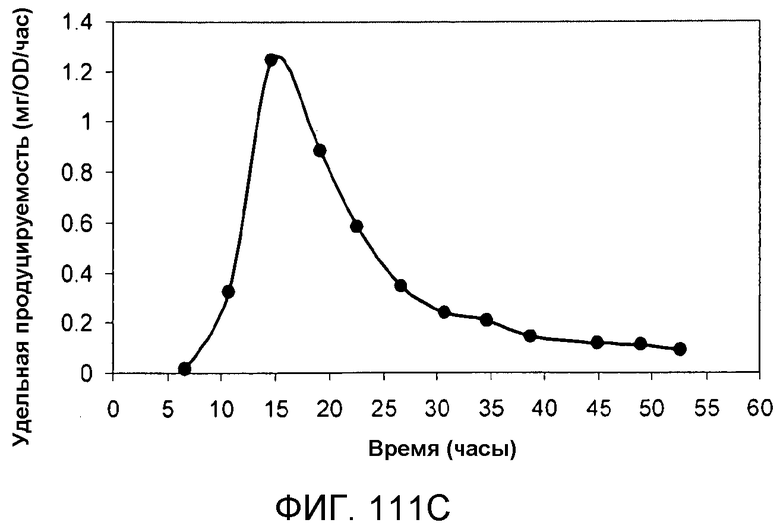
Настоящее изобретение относится к биотехнологии и представляет собой способы продуцирования изопрена культивированием рекомбинантных клеток грибов или микроогранизмов. Указанные клетки обязательно содержат гетерологичный полипептид изопренсинтазы и по меньшей мере одну нуклеиновую кислоту, кодирующую по меньшей мере один полипептид мевалонатного пути (MVA), такой как полипептид ацетил-СоА ацетилтрансферазы, полипептид 3-гидрокси-3-метилглутарил-СоА-синтазы, полипептид 3-гидрокси-3-метилглутарил-СоА-редуктазы, полипептид мевалонаткиназы, полипептид фосфомевалонаткиназы и полипептид дифосфомевалонатдекарбоксилазы. Указанные клетки продуцируют более чем 400 нмоль/гwcm/час изопрена. Настоящее изобретение позволяет получить изопрен методом культивирования указанных рекомбинантных клеток с высоким выходом. 2 н. и 29 з.п. ф-лы, 111 ил., 5 табл., 13 пр.
1. Способ продуцирования изопрена, где указанный способ включает:
(a) культивирование рекомбинантных клеток, содержащих гетерологичный полипептид изопренсинтазы и одну или более нуклеиновых кислот, кодирующих один или более полипептидов мевалонатного пути (MVA), выбранных из группы, состоящей из:
(i) полипептида ацетил-СоА ацетилтрансферазы;
(ii) полипептида 3-гидрокси-3-метилглутарил-СоА-синтазы;
(iii) полипептида 3-гидрокси-3-метилглутарил-СоА-редуктазы;
(iv) полипептида мевалонаткиназы;
(v) полипептида фосфомевалонаткиназы; и
(vi) полипептида дифосфомевалонатдекарбоксилазы,
в условиях культивирования, подходящих для продуцирования изопрена в газовой фазе, и
(b) продуцирование изопрена в газовой фазе, где указанная газовая фаза включает апирогенную концентрацию изопрена и более чем или примерно 9,5% (по объему) кислорода, и где указанные клетки продуцируют более чем примерно 400 нмоль/гwcm/час изопрена.
2. Способ по п.1, где концентрация изопрена в указанной газовой фазе составляет меньше нижнего предела воспламеняемости (LFL) изопрена.
3. Способ по п.2, где LFL изопрена составляет от менее чем примерно 1,5 об.% до примерно 2,0 об.%.
4. Способ по п.1, где концентрация изопрена в указанной газовой фазе составляет больше верхнего предела воспламеняемости (UFL) изопрена.
5. Способ по п.4, где UFL изопрена составляет более чем примерно 12,0 об.%.
6. Способ продуцирования изопрена, где указанный способ включает:
(a) культивирование рекомбинантных клеток, содержащих гетерологичный полипептид изопренсинтазы и одну или более нуклеиновых кислот, кодирующих один или более полипептидов мевалонатного пути (MVA), выбранных из группы, состоящей из:
(i) полипептида ацетил-СоА ацетилтрансферазы;
(ii) полипептида 3-гидрокси-3-метилглутарил-СоА-синтазы;
(iii) полипептида 3-гидрокси-3-метилглутарил-СоА-редуктазы;
(iv) полипептида мевалонаткиназы;
(v) полипептида фосфомевалонаткиназы; и
(vi) полипептида дифосфомевалонатдекарбоксилазы,
в условиях культивирования, подходящих для продуцирования изопрена в газовой фазе, где указанная газовая фаза содержит менее чем или примерно 9,5% (по объему) кислорода, и
(b) продуцирование изопрена в газовой фазе, где указанная газовая фаза включает апирогенную концентрацию изопрена и менее чем или примерно 9,5% (по объему) кислорода, и где указанные клетки продуцируют более чем примерно 400 нмоль/гwcm/час изопрена.
7. Способ по п.6, где концентрация изопрена в указанной газовой фазе составляет меньше LFL изопрена.
8. Способ по п.7, где LFL изопрена составляет от менее чем примерно 1,5 об.% до примерно 2,0 об.%.
9. Способ по п.6, где концентрация изопрена в указанной газовой фазе составляет больше UFL изопрена.
10. Способ по п.9, где UFL изопрена составляет от примерно 2,0 об.% до примерно 12,0 об.%.
11. Способ по п.1 или 6, где изопрен получают в газовой фазе при температуре между приблизительно 25°C и приблизительно 55°C.
12. Способ по п.1 или 6, где изопрен получают в газовой фазе при давлении между приблизительно в одну атмосферу и приблизительно в три атмосферы.
13. Способ по п.1 или 6, где указанный полипептид изопренсинтазы представляет собой полипептид изопренсинтазы растения.
14. Способ по п.1 или 6, где указанные клетки представляют собой бактериальные клетки.
15. Способ по п.14, где указанные бактериальные клетки выбраны из группы, состоящей из клеток Escherichia coli, Pantoae citrea, Bacillus subtilis, Bacillus licheniformis, Bacillus lentus, Bacillus brevis, Bacillus stearothermophilus, Bacillus alkalophilus, Bacillus amyloliquefaciens, Bacillus clausii, Bacillus halodurans, Bacillus megaterium, Bacillus coagulans, Bacillus circulans, Bacillus lautus, Bacillus thuringiensis, Streptomyces lividans, Streptomyces coelicolor, Streptomyces griseus, Pseudomonas sp., и Pseudomonas alcaligenes.
16. Способ по п.1 или 6, где указанные клетки представляют собой клетки грибов.
17. Способ по п.16, где указанные клетки грибов выбраны из группы, состоящей из клеток Aspergillus sp., yeast, Trichoderma sp., Yarrowia sp., Saccharomyces sp., Schizosaccharomyces sp., Pichia sp., Candida sp., Aspergillus oryzae, Aspergillus niger, Saccharomyces cerevisiae, Schizosaccharomyces pombe, Trichoderma reesei, H. insolens, H. lanuginose, H. grisea, Candida lucknowense, Aspergillus sojae, Aspergillus japonicus, Aspergillus nidulans, Aspergillus aculeatus, Aspergillus awamori, Fusarium roseum, Fusarium graminum, Fusarium cerealis, Fusarium oxysporuim, F. venenatum, Neurospora crassa, Mucor miehei, Trichoderma viride, Fusarium oxysporum, и Fusarium solani.
18. Способ по п.1 или 6, где указанные клетки представляют собой клетки водорослей.
19. Способ по п.18, где указанные клетки водорослей выбраны из группы, состоящей из зеленых водорослей, красных водорослей, глаукофитов, хлорарахниофитов, эвгленидов, хромиста и динофлагеллятов.
20. Способ по п.1 или 6, где указанные клетки дополнительно содержат нуклеиновую кислоту, кодирущую полипептид изопентенилдифосфат-дельта-изомеразы (IDI).
21. Способ по п.20, где указанная нуклеиновая кислота, кодирующая полипептид IDI, представляет собой гетерологичную нуклеиновую кислоту.
22. Способ по п.20, где указанный полипептид IDI представляет собой полипептид IDI дрожжей.
23. Способ по п.20, где указанная нуклеиновая кислота, кодирующая полипептид IDI, представляет собой копию эндогенной нуклеиновой кислоты, кодирующей полипептид IDI.
24. Способ по п.1 или 6, где указанные клетки дополнительно содержат гетерологичную нуклеиновую кислоту, кодирущую полипептид 1-дезоксиксилулозо-5-фосфатсинтазы (DXS).
25. Способ по п.1 или 6, где указанные клетки дополнительно содержат копию эндогенной нуклеиновой кислоты, кодирующей полипептид DXS.
26. Способ по п.1 или 6, где по меньшей мере одна из указанных одной или более нуклеиновых кислот, кодирующих один или более полипептидов пути MVA, представляет собой гетерологичную нуклеиновую кислоту.
27. Способ по п.1 или 6, где по меньшей мере одна из указанных одной или более нуклеиновых кислот, кодирующих один или более полипептидов пути MVA, представляет собой копию эндогенной нуклеиновой кислоты.
28. Способ по п.1 или 6, где указанные клетки содержат одну или более нуклеиновых кислот, кодирующих полипептид ацетил-СоА ацетилтрансферазы, полипептид 3-гидрокси-3-метилглутарил-СоА-синтазы, полипептид 3-гидрокси-3-метилглутарил-СоА-редуктазы, полипептид мевалонаткиназы, полипептид фосфомевалонаткиназы и полипептид дифосфомевалонатдекарбоксилазы.
29. Способ по п.1 или 6, где указанные клетки дополнительно содержат:
(a) нуклеиновую кислоту, кодирущую полипептид изопентенилдифосфат-дельта-изомеразы (IDI); и/или
(b) нуклеиновую кислоту, кодирущую полипептид 1-дезоксиксилулозо-5-фосфатсинтазы (DXS).
30. Способ по п.29, где указанная нуклеиновая кислота, кодирующая полипептид DXS, представляет собой гетерологичную нуклеиновую кислоту.
31. Способ по п.29, где указанная нуклеиновая кислота, кодирующая полипептид DXS, представляет собой копию эндогенной нуклеиновой кислоты.
| WO 2008003078 A2, 03.01.2008 | |||
| Пространственный инклиномер | 1978 |
|
SU1118855A1 |
| US 2002095818 A1, 25.07.2002 | |||
| SHARKEY T.D | |||
| et al., Evolution of the isoprene biosynthetic pathway in kudzu, Plant Physyology, February 2005, vol.137, pp.700-712 | |||
| DE 19629568 C1, 08.01.1998 | |||
| KUZMA J | |||
| et al., Bacteria produce the volatile hydrocarbon isoprene, Curr Microbiol, No.30, pp.97-103, February1995, Abstract. | |||
Авторы
Даты
2015-09-20—Публикация
2009-07-01—Подача