Изобретение относится к сельскому хозяйству и может использоваться в селекции и питомниководстве малины.
Малина является ценной ягодной культурой, сладкие и ароматные плоды которой широко используются как в свежем виде, так и для переработки. Малина проста в выращивании, скороплодна, а ягоды ее являются почти первыми в сезоне, когда потребность в плодах особенно велика. В связи с открытием новых антиоксидантных свойств ее плодов в последние два десятилетия заметно вырос спрос на свежие ягоды малины. По производству плодов малины лидирующее место занимает Россия, на долю которой приходится около 30% валового сбора ягод малины в мире (с учетом сбора плодов с дикорастущих насаждений). Благодаря популярности этой культуры научные программы по селекции малины выполняются в нескольких российских селекционных центрах. За последние 5 лет в Государственный реестр селекционных достижений РФ включено 20 новых сортов малины. Все это делает задачу идентификации и паспортизации уже существующих и новых сортов весьма актуальной. Кроме применения для регистрации и сертификации новых селекционных достижений, идентификация сортов также может использоваться и для защиты авторских прав селекционеров в случае нелегального распространения селекционного материала.
В настоящее время для маркирования сортов малины используются как количественные признаки (число побегов, длина междоузлий, длина ягоды), так и качественные (окраска цветка, листьев, побегов и ягод, профиль и морщинистость листьев), которые выявляются визуально в фенотипе растений (RTG/0043/1 «Методика проведения испытаний на отличимость, однородность и стабильность. Малина (Rubus idaeus L.)» от 20.04.2006 г. №12-06/09. http://www.gossort.com/mtd_dus.html). Одним из основных недостатков таких признаков является существенная зависимость их проявления от условий выращивания, а также затрудненная идентификация в определенные сезоны года, например, в отсутствие цветения или плодоношения. В результате заключение о принадлежности данной партии посадочного материала к тому или иному сорту можно сделать лишь с определенной долей вероятности.
В настоящее время более эффективной системой идентификации сортов является молекулярное маркирование. В этой системе в качестве маркеров могут использоваться молекулы белка (изоферментные маркеры) или участки молекулы ДНК (ДНК-маркеры). Идентификация с использованием изоферментных маркеров не требует сложного оборудования, но имеет ряд существенных недостатков. Во-первых, изоферментные маркеры связаны с фенотипом, то есть подвержены влиянию окружающей среды и изменениям на различных стадиях развития растения. Во-вторых, ограниченное количество таких маркеров по причине малочисленности биохимических анализов для их обнаружения не позволяет произвести тщательный анализ генетического разнообразия. В связи с этим, наиболее перспективным является использование в качестве маркерных систем полиморфных нуклеотидных последовательностей ДНК, позволяющих тестировать генетический полиморфизм непосредственно на уровне генов, а не на уровне продуктов генов, как в случае использования метода белкового полиморфизма. Так как последовательность нуклеотидов в молекуле ДНК не зависит от условий внешней среды, то ДНК-маркеры могут использоваться на любой стадии развития организма. Кроме того, эта маркерная система дает возможность использовать для анализа любые ткани и органы, анализировать гербарный материал, длительно хранить образцы в виде выделенной ДНК и т.д.
Методы оценки с помощью ДНК-маркеров делятся на две группы: основанные на гибридизации обработанной рестриктазами ДНК с меченым зондом и на амплификации ДНК со специально выбранными короткими последовательностями (праймерами) методом ПЦР. Методы на основе ПЦР применяются чаще, и среди них широкое распространение получил RAPD-метод, достоинствами которого являются простота и удобство в использовании, невысокая стоимость анализа, потребность в небольшом количестве ДНК и отсутствие необходимости в предварительной информации об исследуемых генотипах.
Известен способ определения генетического разнообразия малины с помощью RAPD-маркеров (Weber С.А. Genetic diversity in black raspberry detected by RAPD markers. HortScience. - 2003. - V. 38. - P. 269-272). Недостатком данного способа является то, что предлагаемый набор праймеров использовался для анализа малины черной (Rubus occidentalis L.). Кроме того, температура отжига при проведении ПЦР для всех праймеров была одинаковой (35°C), что снижает эффективность метода, так как каждый праймер имеет свою, оптимальную для него, температуру отжига.
Наиболее близким к заявляемому изобретению является оценка генетического разнообразия сортов малины красной болгарской селекции с помощью RAPD-маркеров (Badjakov I., Todorovska Е., Kondakova V., Boicheva R., Atanassov A. Assessment the genetic diversity of Bulgarian raspberry germplasm collected by microsatellite and RAPD markers. J. Fruit Ornam. Plant Res. - 2006. - V. 14 (Suppl. 1). - P. 61-76). Недостатком данного способа является малое число используемых праймеров (всего 4), что явно недостаточно для анализа сортов, имеющих родственное происхождение. Кроме того, в реакции ПЦР применялась усредненная для всех праймеров температура отжига (35°C).
Задачей изобретения является повышение уровня идентификации сортов малины на основе ДНК-полиморфизма RAPD-методом за счет увеличения числа используемых RAPD-праймеров.
Поставленная задача решается благодаря тому, что в известном способе идентификации сортов малины на основе RAPD-маркеров, включающем выделение ДНК из растительного материала, амплификацию ДНК в реакционной смеси с последовательным участием нескольких рандомных праймеров, разделение продуктов амплификации с помощью электрофореза, визуализацию продуктов амплификации под ультрафиолетовым светом, выявление для каждого сорта специфичных профилей продуктов амплификации в качестве его маркера, согласно изобретению амплификацию ДНК проводят с использованием увеличенного числа праймеров, имеющих следующую структуру:

причем в процессе амплификации обеспечивают индивидуальную температуру отжига для каждого праймера, при этом выделение ДНК осуществляют из материала, взятого от не менее десяти растений каждого сорта, выращенных в поле или защищенном грунте или не менее двадцати растений каждого сорта, выращенных в условиях in vitro. Кроме того, на основе присущих только для данного сорта профилей составляют генетический паспорт сорта, включающий сочетание локусов и путем сравнения с которым проводят идентификацию сорта малины.
Способ осуществляют следующим образом:
отбирают образцы растительного материала исследуемого сорта, из образцов выделяют ДНК любым известным способом, амплифицируют ДНК в реакционной смеси с последовательным участием указанных нескольких праймеров по известной методике, полученные продукты амплификации разделяют на фракции методом электрофореза в разделяющем геле по стандартной методике, осуществляют визуализацию продуктов амплификации под ультрафиолетовым светом, для каждого сорта выявляют специфичные профили продуктов амплификации, которые служат маркерами данного селекционного достижения, причем в процессе амплификации обеспечивают индивидуальную температуру отжига для каждого праймера. При этом выделение ДНК осуществляют из материала, взятого от не менее десяти растений каждого сорта, выращенных в поле или защищенном грунте или не менее двадцати растений каждого сорта, выращенных в условиях in vitro. Кроме того, на основе присущих только для данного сорта профилей составляют генетический паспорт сорта, включающий сочетание локусов и путем сравнения с которым проводят идентификацию сорта малины.
Примеры осуществления способа
Пример 1. Отбор растительного материала.
Для идентификации сорта отбирали листья с десяти растений для каждого сорта, выращенных в полевых условиях. До начала исследований листья хранили в холодильнике при 4°C. Всего анализировали 18 сортов малины: Агавам, Атлант, Бабье лето II, Беглянка, Бриллиантовая, Брянское диво, Геракл, Желтый гигант, Золотая осень, Кумберленд, Логанберри, Лаутон, Оранжевое чудо, Пингвин, Солнышко, Тайберри, Торнфри, Элегантная.
Пример 2. Выделение ДНК.
Для выделения ДНК использовали модифицированный СТАВ-метод:
Гомогенизация и экстракция. Фрагменты растительной ткани массой до 50 мг помещали в центрифужную пробирку типа "Eppendorf" объемом 1,5 мл, содержащую 500 мкл экстрагирующего буфера (Т=65°C), следующего состава: 1,5% раствор бромида цетилтриметиламмония (СТАВ); 0,12 М раствор трис-HCl (рН 8,0); 1,2 М раствор хлорида натрия; 15 мМ раствор трилона Б, и гомогенизировали до однородного состояния. Далее пробирку закрывали и перемешивали содержимое на вихревом смесителе (400-600 об/мин) в течение 5 секунд. После этого пробирки помещали в твердотельный термостат и инкубировали в течение 30 мин при 65°C.
Очистка гомогенатов. После экстракции пробирку охлаждали до комнатной температуры и к образцу добавляли 500 мкл хлороформа. Содержимое перемешивали на встряхивающей ванне (200 об/мин) в течение 15 мин при комнатной температуре. Далее производили центрифугирование при 13000× g в течение 10 мин. После этого пипеточным дозатором отбирали 300 мкл супернатанта, переносили в другую центрифужную пробирку типа "Eppendorf" объемом 1,5 мл и добавляли к нему 60 мкл буфера 5×СТАВ (5% р-р СТАВ; 350 мМ раствор трилона Б). Содержимое перемешивали на вихревом смесителе (400 об/мин) и инкубировали в твердотельном термостате в течение 10 мин при 65°C. После инкубации добавляли 360 мкл хлороформа. Содержимое перемешивали на встряхивающей ванне (200 об/мин) в течение 10 мин при комнатной температуре, после чего центрифугировали при 13000× g в течение 10 мин (если раствор оставался мутным или была большая интерфаза, то процедуру очистки хлороформом повторяли).
Осаждение ДНК. По окончании центрифугирования пипеточным дозатором отбирали 200 мкл супернатанта, переносили в другую центрифужную пробирку типа "Eppendorf" объемом 1,5 мл и добавляли 200 мкл изопропанола. После добавления спирта содержимое пробирки перемешивали и оставляли в роторе центрифуги на 30 мин. Далее производили центрифугирование при 13000× g (Т=4°C) в течение 10 мин.
Очистка препарата ДНК. Супернатант сливали, а полученный осадок ДНК промывали 700 мкл охлажденного 70% этанола. После промывания содержимое пробирок центрифугировали при 13000× g (Т=4°C) в течение 5 мин. Процедуру промывки повторяли два раза для удаления из осадка остатков трилона Б и изопропанола, поскольку они являются ингибиторами амплификации.
Лиофилизация препарата ДНК. После промывки этанолом, пробирки размещали в штативе и, открыв крышки, просушивали осадок ДНК в течение 30-40 мин (Т=45°C) до полного испарения этанола.
Растворение препарата ДНК. Высушенный осадок растворяли в 50 мкл деионизированной воды во встряхивающей ванне (200 об/мин) при 65°C в течение 15 мин.
Пример 3. Полимеразная цепная реакция (ПЦР).
Для идентификации сортов малины использовали шесть RAPD-праймеров (таблица 1).
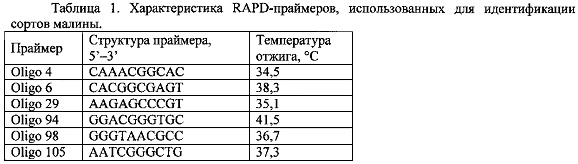
ПЦР проводили по следующей программе:
1 цикл - длительная денатурация (3 мин, 94°C); 40 циклов - денатурация (30 с, 94°C), отжиг (30 с, температура отжига из табл. 1), элонгация (45 с, 72°C); 1 цикл - длительная элонгация (7 мин, 72°C).
Пример 4. Электрофоретическое разделение, визуализация и интерпретация результатов ПЦР.
Электрофоретическое разделение продуктов ПЦР проводили в 1,5% агарозных гелях, которые приготавливали путем растворения 1,5 г агарозы в 98,5 г 1× трис-ЭДТА-боратного буфера. Гели помещали в электрофоретическую камеру типа "SE-2" (фирма "Helicon"). Катодный и анодный отсеки заполняли 1× трис-ЭДТА-боратным буфером так, чтобы гель был погружен в буфер на 5 мм. Загрузочный раствор каждой дорожки состоял из 5 мкл продуктов ПЦР и 2 мкл загрузочного буфера (30% глицерин, 0,5% бромфеноловый синий). Электрофорез проводили при 4-8°C в течение 2,0-2,5 часов при параметрах тока 90 V/60 мА.
Визуализацию электрофоретических спектров осуществляли путем помещения гелевой пластины в раствор бромистого этидия (0,5 мкг/мл) на 15 мин. Затем гель извлекали, промывали в дистиллированной воде для удаления остатков красителя и просматривали в УФ-свете. Фракции ДНК проявляются в виде светлых полос на темном фоне.
Интерпретацию полученных результатов проводили визуально. RAPD-маркеры характеризуются доминантным типом проявления и диаллельной системой: "1" - наличие ампликона (доминантный аллель) и "0" - отсутствие ампликона (рецессивный аллель), в той или иной зоне гелевой пластины.
Для составления генетических паспортов учитывали фракции ДНК, воспроизводившиеся у всех десяти (двадцати) растений для каждого сорта.
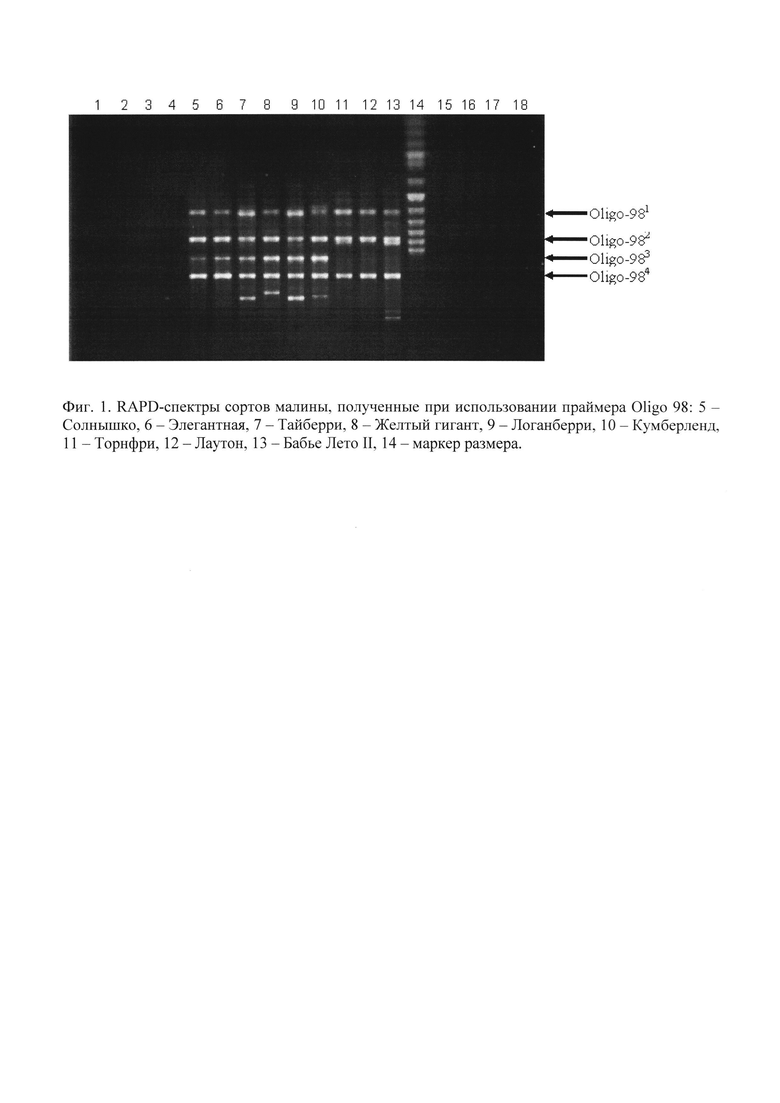
На Фиг. 1 на примере девяти сортов малины представлены RAPD-спектры, полученные при использовании праймера Oligo 98.
Пример 5. Генотипирование различных сортов малины.
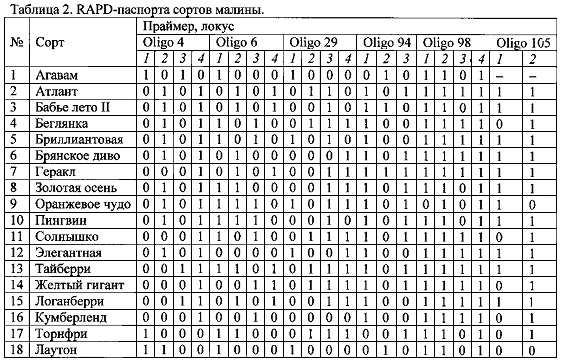
На основании RAPD-спектров, полученных с использованием шести праймеров (Oligo 4, 6, 29, 94, 98, 105), составили многолокусные генетические паспорта различных сортов малины (таблица 2). Каждый из сортов характеризуется определенным уникальным сочетанием локусов. Это сочетание служит эталоном при сравнении и идентификации растений неизвестных сортов малины. Путем сравнения сочетания локусов исследуемых и эталонных образцов определяют сортовую принадлежность малины.
Таким образом, предложенный способ, включающий отбор растительного материала, выделение ДНК из отобранного материала, генетический анализ ДНК RAPD-методом с последовательным участием нескольких праймеров, с последующим разделением продуктов амплификации путем электрофореза, их визуализацией под ультрафиолетовым светом, выявлением специфичных профилей и построение на их основе генетического паспорта, позволит проводить идентификацию любого сорта малины, выращиваемого в России.
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ генетической паспортизации селекционных достижений малины на основе RAPD-маркеров | 2017 |
|
RU2671678C2 |
| СПОСОБ МОЛЕКУЛЯРНОГО МАРКИРОВАНИЯ, ОСНОВАННЫЙ НА МИКРОСАТЕЛЛИТНЫХ ЛОКУСАХ, ПРЕДНАЗНАЧЕННЫЙ ДЛЯ ГЕНЕТИЧЕСКОЙ ПАСПОРТИЗАЦИИ СЕЛЕКЦИОННЫХ ДОСТИЖЕНИЙ РАСТЕНИЙ РОДА RUBUS | 2018 |
|
RU2732922C2 |
| Способ генетической паспортизации сортов розы эфиромасличной с использованием ISSR-маркеров | 2023 |
|
RU2802945C1 |
| СПОСОБ ДНК-ИДЕНТИФИКАЦИИ И ГЕНЕТИЧЕСКОЙ ПАСПОРТИЗАЦИИ СОРТОВ РАЙГРАСА ПАСТБИЩНОГО И ОДНОЛЕТНЕГО НА ОСНОВЕ СИСТЕМ SSR- и SCoT-МАРКИРОВАНИЯ | 2023 |
|
RU2826148C1 |
| БИОЛОГИЧЕСКИЙ ДНК МАРКЕР ДЛЯ ОПРЕДЕЛЕНИЯ СОРТОВ КАРТОФЕЛЯ, НАБОР И СПОСОБ СОРТОВОЙ ИДЕНТИФИКАЦИИ КАРТОФЕЛЯ | 2009 |
|
RU2413774C1 |
| СПОСОБ СЕЛЕКЦИИ ГЕНОТИПОВ МАЛИНЫ НА СОДЕРЖАНИЕ АНТОЦИАНОВ С ПОМОЩЬЮ МОЛЕКУЛЯРНЫХ МАРКЕРОВ | 2019 |
|
RU2750958C2 |
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОЙ ИДЕНТИФИКАЦИИ СОРТОВ И ЛИНИЙ СОИ | 2024 |
|
RU2839697C1 |
| ДНК-маркер для селекции сортов ярового ячменя, устойчивых к темно-бурой пятнистости | 2020 |
|
RU2740404C1 |
| СПОСОБ ИДЕНТИФИКАЦИИ СОРТОВ СОИ НА ОСНОВЕ МИКРОСАТЕЛЛИТНЫХ (SSR) МАРКЕРОВ | 2008 |
|
RU2388828C1 |
| НОВЫЙ ТИП ГЕНЕТИЧЕСКОГО МАРКЕРА НА ОСНОВЕ ТРАНСПОЗОНА | 2000 |
|
RU2279482C2 |
Изобретение относится к области биохимии, в частности к способу идентификации сортов малины Агавам, Атлант, Бабье лето II, Беглянка, Бриллиантовая, Брянское диво, Геракл, Желтый гигант, Золотая осень, Кумберленд, Логанберри, Лаутон, Оранжевое чудо, Пингвин, Солнышко, Тайберри, Торнфри, Элегантная на основе RAPD-маркеров, включающий отбор растительного материала, выделение ДНК из растительного материала, амплификацию ДНК в реакционной смеси с последовательным участием нескольких рандомных праймеров, разделение продуктов амплификации с помощью электрофореза, визуализацию продуктов амплификации под ультрафиолетовым светом и выявление среди продуктов амплификации уникальных сочетаний аллелей всех амплифицируемых зон (локусов) по всем используемым праймерам, присущих только для данного сорта профилей. При этом амплификацию ДНК проводят с последовательным участием следующих рандомных праймеров: Oligo 4 - 5'-CAAACGGCAC-3', Oligo 6 - 5'-CACGGCGAGT-3', Oligo 29 - 5'-AAGAGCCCGT-3', Oligo 94 - 5'-GGACGGGTGC-3', Oligo 98 - 5'-GGGTAACGCC-3', Oligo 105 - 5' -AATCGGGCTG-3'. Изобретение позволяет эффективно идентифицировать сорта малины. 3 з.п. ф-лы, 1 ил., 2 табл., 5 пр.
1. Способ идентификации сортов малины Агавам, Атлант, Бабье лето II, Беглянка, Бриллиантовая, Брянское диво, Геракл, Желтый гигант, Золотая осень, Кумберленд, Логанберри, Лаутон, Оранжевое чудо, Пингвин, Солнышко, Тайберри, Торнфри, Элегантная на основе RAPD-маркеров, включающий отбор растительного материала, выделение ДНК из растительного материала, амплификацию ДНК в реакционной смеси с последовательным участием нескольких рандомных праймеров, разделение продуктов амплификации с помощью электрофореза, визуализацию продуктов амплификации под ультрафиолетовым светом и выявление среди продуктов амплификации уникальных сочетаний аллелей всех амплифицируемых зон (локусов) по всем используемым праймерам, присущих только для данного сорта профилей:
Агавам: Oligo 4 - локус 1аллель 1, локус 2аллель 0, локус 3аллель 1, локус 4 аллель 0; Oligo 6 - 11,20,30,40; Oligo 29 - 11;20;30;40; Oligo 94 - 10;21;30; Oligo 98 - 11;21;30;41; Oligo 105 - 1-;2-;
Атлант: 4 - 10,21,30,41; 6 - 10,21,30,41; 29 - 10,21,31,40; 94 - 11,20,31; 98 - 11,21,31,41; 105 - 11,21;
Бабье лето II: 4 - 10,21,30,41; 6 - 10,21,30,41; 29 - 10,20,31,40; 94 - 11,21,30; 98 - 11,21,30,41; 105 - 11,21;
Беглянка: 4 - 10,21,30,41; 6 - 11,20,31,40; 29 - 10,21,31,41; 94 - 11,20,30; 98 - 11,21,31,41; 105 - 10,21;
Бриллиантовая: 4 - 10,21,30,41; 6 - 11,20,31,40; 29 - 11,20,31,40; 94 - 11,20,30; 98 - 11,21,31,41; 105 - 11,21;
Брянское диво: 4 - 10,21,30,41; 6 - 10,21,30,40; 29 - 10,20,30,41; 94 - 11,20,31; 98 - 11,21,31,41; 105 - 11,21;
Геракл: 4 - 10,20,30,41; 6 - 10,21,30,41; 29 - 10,20,31,41; 94 - 11,21,31; 98 - 11,21,31,41; 105 - 11,21;
Золотая осень: 4 - 10,21,30,41; 6 - 11,21,30,40; 29 - 10,20,31,41; 94 - 11,20,31; 98 - 11,21,30,41; 105 - 11,21;
Оранжевое чудо: 4 - 10,21,30,41; 6 - 11,21,31,40; 29 - 11,20,31,41; 94 - 11,20,31; 98 - 10,21,30,41; 105 - 11,20;
Пингвин: 4 - 10,21,30,41; 6 - 11,21,31,40; 29 - 10,20,31,40; 94 - 11,20,31; 98 - 11,21,30,41; 105 - 11,21;
Солнышко: 4 - 10,20,30,41; 6 - 11,20,31,40; 29 - 10,21,31,41; 94 - 11,20,31; 98 - 11,21,31,41; 105 - 10,21;
Элегантная: 4 - 10,21,30,41; 6 - 10,20,30,40; 29 - 11,20,30,41; 94 - 11,20,30; 98 - 11,21,31,41; 105 - 11,21;
Тайберри: 4 - 10,20,31,41; 6 - 11,21,30,41; 29 - 10,21,31,41; 94 - 11,20,31; 98 - 11,21,31,41; 105 - 11,21;
Желтый гигант: 4 - 10,20,30,41; 6 - 10,20,31,41; 29 - 10,21,31,41; 94 - 11,20,31; 98 - 11,21,31,41; 105 - 10,21;
Логанберри: 4 - 10,20,31,41; 6 - 10,21,30,41; 29 - 10,20,31,41; 94 - 11,20,30; 98 - 11,21,31,41; 105 - 11,21;
Кумберленд: 4 - 10,20,30,41; 6 - 10,20,31,40; 29 - 10,20,30,40; 94 - 11,20,30; 98 - 11,21,31,41; 105 - 10,21;
Торнфри: 4 - 11,20,30,40; 6 - 11,21,30,40; 29 - 10,21,31,41; 94 - 10,20,31; 98 - 11,21,30,41; 105 - 10,21;
Лаутон: 4 - 11,21,30,40; 6 - 11,20,30,40; 29 - 11,20,30,40; 94 - 10,21,30; 98 - 11,21,30,41; 105 - 10,20,
отличающийся тем, что амплификацию ДНК проводят с последовательным участием следующих рандомных праймеров:
Oligo 4 - 5'-CAAACGGCAC-3'
Oligo 6 - 5'-CACGGCGAGT-3'
Oligo 29 - 5'-AAGAGCCCGT-3'
Oligo 94 - 5'-GGACGGGTGC-3'
Oligo 98 - 5'-GGGTAACGCC-3'
Oligo 105 - 5' -AATCGGGCTG-3'
2. Способ по п. l, отличающийся тем, что для каждого рандомного праймера обеспечивают индивидуальную температуру отжига (°С):
Oligo 4 - 34,5
Oligo 6 - 38,3
Oligo 29-35,1
Oligo 94-41,5
Oligo 98 - 36,7
Oligo 105 - 37,3
3. Способ по п. 1, отличающийся тем, что выделение ДНК осуществляют из материала, взятого от не менее десяти растений каждого сорта, выращенных в поле или защищенном грунте или не менее двадцати растений каждого сорта, выращенных в условиях in vitro.
4. Способ по п. 1, отличающийся тем, что на основе присущих только для данного сорта профилей составляют генетический паспорт сорта, включающий сочетание локусов, и путем сравнения с которым проводят идентификацию сорта малины.
| BADJAKOV I | |||
| et al., Assessment the genetic diversity of Bulgarian raspberry germplasm collected by microsatellite and RAPD markers, J | |||
| Fruit Ornam | |||
| Plant Res, 2006, V | |||
| Паровоз для отопления неспекающейся каменноугольной мелочью | 1916 |
|
SU14A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Устройство для сортировки каменного угля | 1921 |
|
SU61A1 |
| КАГАН Д.И | |||
| и др., ПАСПОРТИЗАЦИЯ СОРТОВ МАЛИНЫ И ЕЖЕВИКИ И ИЗУЧЕНИЕ ИХ ФИЛОГЕНЕТИЧЕСКИХ ВЗАИМООТНОШЕНИЙ МЕТОДОМ RAPD-АНАЛИЗА, Биотехнологические приемы в сохранении биоразнообразия и селекции растений | |||
| Сборник статей Международной научной конференции, Минск, 18-20 августа 2014 г | |||
| WEBER С.А., Genetic diversity in black raspberry detected by RAPD markers, HortScience, 2003, Vol | |||
| Способ сужения чугунных изделий | 1922 |
|
SU38A1 |
| Нож для надрезывания подошвы рантовой обуви | 1917 |
|
SU269A1 |
| СПОСОБ МАРКИРОВАНИЯ СЕЛЕКЦИОННЫХ ДОСТИЖЕНИЙ КЛЕВЕРА ЛУГОВОГО НА ОСНОВЕ RAPD-МАРКЕРОВ | 2002 |
|
RU2244416C2 |
Авторы
Даты
2017-05-30—Публикация
2015-02-17—Подача