Область изобретения
Изобретение относится к области генетики человека, более конкретно, к способу конструирования единичного олигонуклеотида, который предпочтительно способен индуцировать пропускание двух или более экзонов пре-мРНК. Кроме того, изобретение относится к указанному олигонуклеотиду, к фармацевтической композиции, содержащей указанный олигонуклеотид, и к применению указанного олигонуклеотида, как указано в настоящем описании.
Уровень техники, к которому относится изобретение
Олигонуклеотиды являются перспективными в медицине для лечения генетических нарушений, таких как мышечная дистрофия. Мышечная дистрофия (MD) относится к генетическим заболеваниям, которые характеризуются прогрессирующей слабостью и дегенерацией скелетных мышц. Мышечная дистрофия Дюшенна (DMD) и мышечная дистрофия Беккера (BMD) являются наиболее распространенными детскими формами мышечной дистрофии, и они используются в настоящем описании для иллюстрации изобретения. DMD представляет собой тяжелое летальное нервно-мышечное нарушение, приводящее к потребности в инвалидной коляске в возрасте до 12 лет и пациенты DMD часто погибают в возрасте до тридцати лет вследствие дыхательной или сердечной недостаточности.
DMD вызывается мутациями в гене DMD; в основном делециями или дупликациями со сдвигом рамки считывания одного или нескольких экзонов, небольшими нуклеотидными вставками или делециями, или точечными нонсенс-мутациями, которые, как правило, приводят к отсутствию функционального дистрофина. В ходе последнего десятилетия появилась специально индуцируемая модификация сплайсинга для восстановления нарушенной рамки считывания транскрипта DMD в качестве перспективной терапии мышечной дистрофии Дюшенна (DMD) (van Ommen G.J. et al., Yokota T., et al., van Deutekom et al., Goemans N.M., et al.). С использованием специфичных к последовательности антисмысловых олигонуклеотидов (AON), которые нацелены на конкретный экзон, фланкирующий или содержащий мутацию, и препятствуют его сигналам сплайсинга, может быть индуцировано пропускание этого экзона во время процессинга пре-мРНК DMD. Несмотря на получаемый в результате укороченный транскрипт, открытая рамка считывания восстанавливается, и образуется белок, который сходен с белками, встречающимися у пациентов с обычно более мягкой мышечной дистрофией Беккера. Индуцируемое AON пропускание экзонов обеспечивает специфический для мутации и, таким образом, потенциально персонализированный терапевтический подход для пациентов с DMD и определенных пациентов с тяжелой BMD. Поскольку большинство мутаций кластеризуются в области экзонов 45-55 в гене DMD, пропускание одного конкретного экзона в этой области может быть терапевтическим для субпопуляции пациентов с различными мутациями. Пропускание экзона 51 имеют наибольшие субпопуляции пациентов (приблизительно 13%), включая пациентов с делециями экзонов 45-50, 48-50, 50 или 52. В случае некоторых мутаций для восстановления открытой рамки считывания требуется пропускание более одного экзона. Например, для пациентов DMD с делецией с экзона 46 по экзон 50 в гене DMD, только пропускание обоих из экзонов 45 и 51 является корректирующим. Для лечения этих пациентов требуется введение двух олигонуклеотидов, один из которых нацелен на экзон 45, а другой из которых нацелен на экзон 51. Осуществимость пропускания двух или более последовательных экзонов с использованием комбинации AON, либо в коктейле, либо в доставляемых с помощью вирусов генных конструкциях, широко исследована (Aartsma-Rus A. et al., 2004; Béroud C., et al.; Van Vliet L., et al.; Yokota T., et al.; Goyenvalle A., et al.). Пропускание множества экзонов было бы применимым для совокупных субпопуляций пациентов, позволило бы имитировать делеции, о которых известно, что они ассоциированы с относительно мягкими фенотипами, и обеспечило бы инструмент для нацеливания на редкие мутации вне области горячей точки делеций в гене DMD. Однако недостатком разработки лекарственных средств, содержащих множество олигонуклеотидов, является то, что учреждение по контролю за оборотом лекарственных средств может рассматривать олигонуклеотиды с различными последовательностями как различные лекарственные средства, для каждого из которых требуются доказательства стабильной продукции, испытания токсичности и клинические испытания. Таким образом, существует потребность в одном молекулярном соединении, способном индуцировать пропускание по меньшей мере двух экзонов, чтобы упростить лечение совокупных подгрупп пациентов с DMD.
Описание изобретения
Изобретение относится к способу конструирования олигонуклеотида, где указанный олигонуклеотид способен связываться с областью первого экзона и с областью второго экзона в одной и той же пре-мРНК, где указанная область указанного второго экзона обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона. Олигонуклеотиды, получаемые указанным способом, предпочтительно способны индуцировать пропускание указанного первого экзона и указанного второго экзона в указанной пре-мРНК. Предпочтительно, также индуцируется пропускание дополнительного экзона(ов), где указанный дополнительный экзон(ы) предпочтительно расположен между указанным первым и указанным вторым экзонами. Полученный транскрипт указанной пре-мРНК, где пропускаются указанные экзоны, находится в рамке считывания.
Олигонуклеотид
В первом аспекте изобретение относится к олигонуклеотиду, который способен связываться с областью первого экзона и с областью второго экзона в одной и той же пре-мРНК, где указанная область второго экзона обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона.
Олигонуклеотид предпочтительно способен индуцировать пропускание первого и второго экзонов указанной пре-мРНК; более предпочтительно, индуцируется пропускание дополнительного экзона(ов), где указанный дополнительный экзон(ы) предпочтительно расположен между указанным первым и указанным вторым экзонами, и где полученный транскрипт находится в рамке считывания.
Пропускание экзона препятствует природным процессам сплайсинга, происходящим в эукариотической клетке. У высших эукариот генетическая информация для белков в ДНК клетки кодируется в экзонах, которые отделены друг от друга последовательностями интронов. Эти интроны в некоторых случаях являются очень длинными. Аппарат транскрипции эукариот образует пре-мРНК, которая содержит как экзоны, так и интроны, в то время как аппарат сплайсинга, часто уже во время происходящей продукции пре-мРНК, образует истинную кодирующую мРНК для белка путем удаления интронов и связывания экзонов, присутствующих в пре-мРНК, в ходе процесса, называемого сплайсингом.
Олигонуклеотид по изобретению, который способен связываться с областью первого экзона в пре-мРНК и с областью второго экзона в той же пре-мРНК, следует рассматривать как олигонуклеотид, пригодный для связывания с областью первого экзона в пре-мРНК и пригодный для связывания с областью второго экзона в той же пре-мРНК. Такой олигонуклеотид по изобретению характеризуется его признаком связывания (т.е. способен связываться), когда его используют с или в комбинации с пре-мРНК, предпочтительно в клетке. В этом контексте выражение "способен к" может быть заменено на "может". Таким образом, специалисту в данной области будет понятно, что олигонуклеотид, способный связываться с областью первого экзона и способный связываться с областью второго экзона в одной и той же пре-мРНК, определяемый нуклеотидной последовательностью, определяет указанный олигонуклеотид структурно, т.е. указанный олигонуклеотид обладает такой последовательностью, которая обратно комплементарна последовательности указанной области указанного первого экзона и также обратно комплементарна последовательности указанной области указанного второго экзона в одной и той же пре-мРНК. Степень обратной комплементарности с указанными областями указанного первого и/или указанного второго экзона, которая необходима для олигонуклеотида по изобретению, может составлять менее 100%. Может быть допустимым определенное количество несоответствий нуклеотидов или один или два пропуска, как дополнительно описано в настоящем описании. Нуклеотидную последовательность области первого экзона, которая обладает по меньшей мере 50% идентичностью с указанной областью указанного второго экзона (или нуклеотидная последовательность области второго экзона, которая обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона), с которой способен связываться олигонуклеотид по изобретению, можно конструировать с использованием способа по изобретению, как пояснено ниже в настоящем описании. Предпочтительная пре-мРНК представляет собой пре-мРНК дистрофина. Предпочтительные комбинации первого и второго экзонов пре-мРНК дистрофина и предпочтительные области указанных первого и второго экзонов дистрофина определены в таблице 2. Олигонуклеотид по изобретению предпочтительно способен индуцировать пропускание указанных первого и второго экзонов в указанной пре-мРНК дистрофина; более предпочтительно, индуцируется пропускание дополнительного экзона(ов), где указанный дополнительный экзон(ы) предпочтительно расположен между указанным первым и указанным вторым экзонами, и где полученный транскрипт дистрофина находится в рамке считывания, предпочтительно, как указано в таблице 1.
Транскрипт находится в рамке считывания, когда он имеет открытую рамку считывания, которая обеспечивает продукцию белка. Состояние нахождения мРНК в рамке считывания можно оценивать с помощью анализа последовательности и/или анализа ОТ-ПЦР, как известно специалисту в данной области. Полученный белок, который образуется в результате трансляции транскрипта в рамке считывания, можно анализировать с помощью иммунофлуоресценции и/или анализа с использованием вестерн-блоттинга с использованием антител, которые перекрестно реагируют с указанным белком, как известно специалисту в данной области. В рамках настоящего изобретения олигонуклеотид, описанный в настоящем описании, может быть назван функциональным, если полученный транскрипт в рамке считывания идентифицируется с помощью ОТ-ПЦР и/или анализа последовательности, или если белок, образовавшийся с указанного транскрипта, идентифицируется с помощью иммунофлуоресценции и/или анализа с использованием вестерн-блоттинга, в соответствующей системе in vitro или in vivo в зависимости от типа транскрипта. Если транскрипт представляет собой транскрипт дистрофина, соответствующая система может представлять собой мышечную клетку или мышечную трубочку здорового донора или пациента с DMD, как пояснено в настоящем описании ниже.
В одном варианте осуществления область второго экзона (присутствующая в той же пре-мРНК в качестве области первого экзона) обладает по меньшей мере 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% или 100% идентичностью с областью первого экзона, как определено выше (предпочтительные области первого и второго экзонов дистрофина указаны в таблице 2). Процент идентичности можно оценивать по всей длине указанных первого и/или второго экзонов или на протяжении области из 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200 или более нуклеотидов, как проиллюстрировано в настоящем описании для экзонов дистрофина. Для квалифицированного специалиста очевидно, что первый и второй экзоны, как описано в настоящем описании, представляют собой два различных экзона одной единственной мРНК или два различных экзона одной и той же пре-мРНК. Первый экзон, как указано в настоящем описании, может быть расположен выше (т.е. с 5'-стороны) второго экзона в одной и той же пре-мРНК, как указано в настоящем описании, или второй экзон может быть расположен выше указанного первого экзона. Предпочтительно, указанный первый экзон расположен выше указанного второго экзона. Специалисту в данной области очевидно, что олигонуклеотид по изобретению может быть исходно сконструирован так, чтобы он был способен связываться с областью первого экзона; ввиду идентичности области указанного первого и указанного второй экзонов, указанный олигонуклеотид также вторично может быть способен связываться с указанной областью указанного второго экзона. Возможно обратное конструирование: олигонуклеотид по изобретению может быть первично сконструирован так, чтобы он был способен связываться с областью второго экзона; ввиду идентичности области указанного первого и указанного второго экзонов, указанный олигонуклеотид также может быть вторично способен связываться с указанной областью указанного первого экзона.
Процент идентичности между областью первого и областью второго экзонов можно оценивать по всей области указанного первого экзона, где область может быть более короткой, более длинной или в равной степени длинной относительно части этой области, с которой олигонуклеотид по изобретению способен связываться. Область первого экзона и область второго экзона, как используют в рамках изобретения, также могут быть идентифицированы как область(и) идентичности. Предпочтительно область первого экзона, которая определяет идентичность с областью второго экзона, является в равной степени длинной или более длинной, чем часть этой области, с которой способен связываться олигонуклеотид по изобретению. Должно быть понятно, что олигонуклеотид по изобретению может быть способен связываться с меньшей частью или частично перекрывающейся частью указанных областей, использованных для оценки идентичности последовательностей указанных первого и/или второго экзонов. Таким образом, должно быть понятно, что олигонуклеотид, который способен связываться с областью первого и второго экзонов, может связываться с частью указанной области указанного первого экзона и указанного второго экзона. Указанная часть может иметь такую же длину, как и указанная область указанного первого и/или указанного второго экзонов. Указанная часть может быть более короткой или более длинной относительно указанной области указанного первого и/или указанного второго экзонов. Указанная часть может находиться в указанной области указанного первого и/или указанного второго экзонов. Указанная часть может перекрываться с указанной областью указанного первого и/или указанного второго экзонов. Это перекрывание может составлять 1, 2, 3, 4, 5 или более нуклеотидов на 5'- и/или на 3'-стороне области указанного первого и/или второго экзона. Олигонуклеотид может быть по меньшей мере на 1, 2, 3, 4, 5 или более нуклеотидов длиннее или короче области указанного первого и/или указанного второго экзона и может находиться на 5'- или 3'-стороне указанной области первой и/или второй области.
Область, составляющая 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80 или вплоть до 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200 или более нуклеотидов, используемая для вычисления процента идентичности между первым экзоном и вторым экзоном, может представлять собой непрерывный участок, или она может прерываться одним, двумя, тремя, четырьмя или более пропусками при условии, что процент идентичности на протяжении всей области составляет по меньшей мере 50%.
Процент идентичности между областью первого экзона и областью второго экзона можно оценивать с использованием любой программы, известной специалисту в данной области. Предпочтительно, указанную идентичность оценивают следующим образом: наилучшее попарное выравнивание между первым и вторым экзонами с использованием сетевого инструмента EMBOSS Matcher с использованием параметров по умолчанию (матрица: EDNAFULL, штраф за внесение пропуска: 16, штраф за продолжение пропуска: 4).
Олигонуклеотид, как используют в рамках изобретения, предпочтительно относится к олигомеру, который способен связываться с, нацеливаться на, гибридизоваться с и/или является обратно комплементарным области или части области первого и второго экзонов в одной и той же пре-мРНК.
Олигонуклеотид, как определено в настоящем описании (т.е. который способен связываться с областью первого экзона и с областью другого экзона (т.е. второго экзона) в одной и той же пре-мРНК, где указанная область указанного второго экзона обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона), также предпочтительно по меньшей мере на 80% обратно комплементарен указанной области указанного первого экзона и по меньшей мере на 45% обратно комплементарен указанной области указанного второго экзона. Более предпочтительно, указанный олигонуклеотид по меньшей мере на 85%, 90%, 95% или 100% обратно комплементарен указанной области указанного первого экзона и по меньшей мере на 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% или 100% обратно комплементарен указанной области указанного второго экзона. Обратную комплементарность предпочтительно, но не обязательно, оценивают на протяжении всей длины олигонуклеотида.
Олигонуклеотид, охватываемый изобретением, может содержать по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39 или 40 нуклеотидов. Олигонуклеотид, охватываемый изобретением, может содержать не более 40, 39, 38, 37, 36, 35, 34, 33, 32, 31, 30, 29, 28, 27, 26, 25, 24, 23, 22, 21, 20, 19, 18, 17, 16, 15, 14, 13, 12, 11, 10 нуклеотидов. Длина олигонуклеотида по изобретению определяется общим количеством нуклеотидов, охватываемых указанным олигонуклеотидом, независимо от модификаций, присутствующих в указанном олигонуклеотиде. Как дополнительно рассмотрено ниже, нуклеотиды могут содержать определенные химические модификации, однако такие модифицированные нуклеотиды, тем не менее, считаются нуклеотидами в контексте настоящего изобретения. В зависимости от химии олигонуклеотида, оптимальная длина олигонуклеотида может отличаться. Например, длина 2'-O-метилфосфоротиоатного олигонуклеотида может составлять от 15 до 30. Если этот олигонуклеотид далее модифицировать, как проиллюстрировано в настоящем описании, оптимальная длина может быть укорочена до 14, 13 или даже менее.
В предпочтительном варианте осуществления олигонуклеотид по изобретению имеет не более 30 нуклеотидов для ограничения вероятности снижения эффективности синтеза, выхода, частоты или масштабируемости, снижения биодоступности и/или клеточного захвата и транспорта, снижения безопасности и для ограничения расходов. В более предпочтительном варианте осуществления олигонуклеотид состоит из 15-25 нуклеотидов. Наиболее предпочтительно, олигонуклеотид, охватываемый изобретением, состоит из 20, 21, 22, 23, 24 или 25 нуклеотидов. Длина олигонуклеотида по изобретению предпочтительно является такой, что функциональность или активность олигонуклеотида определяется индукцией по меньшей мере 5% пропускания первого и второго экзонов (и любого экзона(ов) между ними), или способствованием тому, что образуется по меньшей мере 5% транскрипта в рамке считывания, когда по меньшей мере 100 нМ указанного олигонуклеотида используют для трансфицирования соответствующей клеточной культуры in vitro. Оценка присутствия указанного транскрипта уже описана в настоящем описании. Соответствующая клеточная культура представляет собой клеточную культуру, где пре-мРНК, содержащая указанный первый и указанный второй экзоны, транскрибируется и сплайсируется в мРНК-транскрипт. Если пре-мРНК представляет собой пре-мРНК дистрофина, соответствующая клеточная культура содержит (дифференцированные) мышечные клетки. В этом случае образуется по меньшей мере 20% транскрипта в рамке считывания, когда используют по меньшей мере 250 нМ указанного олигонуклеотида.
Область первого экзона может иметь длину по меньшей мере 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80 или вплоть до 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200 или более нуклеотидов. Область первого экзона также может быть определена как составляющая по меньшей мере 1%, 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или 100% длины указанного экзона. Область первого экзона может быть названа областью идентичности.
Область второго экзона может составлять по меньшей мере 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80 или вплоть до 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200 или более нуклеотидов. Область второго экзона может быть определена как составляющая по меньшей мере 1%, 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или 100% от длины указанного экзона. Область второго экзона может быть названа областью идентичности.
В одном варианте осуществления олигонуклеотид по изобретению способен связываться с областью экзона U+1 (первый экзон) из пре-мРНК, где область другого экзона D-1 (второй экзон) в той же пре-мРНК обладает по меньшей мере 50% идентичностью с указанной областью указанного (U+1) экзона, где указанный олигонуклеотид предназначен для пропускания указанного U+1 и указанного D-1 экзонов (и дополнительного экзона(ов), предпочтительно расположенного между указанным первым и указанным вторым экзонами) указанной пре-мРНК, с образованием транскрипта в рамке считывания, в котором экзоны U и D сплайсированы вместе (например, для DMD, предпочтительно, как в таблице 1). Олигонуклеотид по изобретению также обозначается в настоящем описании как соединение. Олигонуклеотид по изобретению предпочтительно представляет собой антисмысловой олигонуклеотид (т.е. AON). Олигонуклеотид предпочтительно предназначен для пропускания указанных двух экзонов (т.е. указанного первого (U+1) и указанного второго (D-1) экзонов) указанной пре-мРНК, и где образовавшийся транскрипт (в котором U прямо сплайсирован с D) находится в рамке считывания (например, для DMD, предпочтительно, как в таблице 1). Можно сказать, что указанный олигонуклеотид индуцирует пропускание указанных двух экзонов в одной единственной пре-мРНК. Необязательно, индуцируется пропускание дополнительного экзона(ов), где указанный дополнительный экзон(ы) предпочтительно расположен между указанным первым и указанным вторым экзонами, и образующийся транскрипт находится в рамке считывания.
Более предпочтительно, олигонуклеотид предназначен для пропускания указанных двух экзонов (т.е. указанного первого и указанного второго экзонов), и полного участка экзонов между указанным первым и указанным вторым экзонами в указанной пре-мРНК для удаления какой-либо мутации в указанном участке и для получения транскрипта, который является более коротким, но который обладает восстановленной открытой рамкой считывания, что обеспечивает продуцирование белка.
Без связи с какой-либо теорией, полагают, что вследствие по меньшей мере 50% идентичности или сходства последовательностей между указанными двумя экзонами, единственный олигонуклеотид по изобретению способен связываться и индуцировать пропускание обоих экзонов и предпочтительно всего участка экзонов между ними, чтобы получить более короткий транскрипт, который находится в рамке считывания. Таким образом, указанные два экзона могут быть соседними в пре-мРНК, или они могут быть разделены по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49 или 50 экзонами. Область, охватывающая один или несколько экзонов, присутствующих между указанным первым и указанным вторым экзонами, также может быть названа участком (множества) экзонов или мультиэкзонным участком. Предпочтительно, первый экзон этого мультиэкзонного участка представляет собой первый экзон, определенный в настоящем описании выше, а последний экзон этого мультиэкзонного участка представляет собой второй экзон, определенный в настоящем описании ранее. Олигонуклеотид по изобретению также может быть определен как олигонуклеотид, который способен индуцировать пропускание указанных двух экзонов, или пропускание участка (множества) экзонов или пропускание указанного мультиэкзонного участка. В предпочтительном варианте осуществления пропускание как первого экзона, так и второго экзона индуцируют с использованием одного единственного олигонуклеотида по изобретению. В предпочтительном варианте осуществления осуществляют пропускание более одного, более 2, более 3, более 4, более 5, более 6, более 7, более 8, более 9, более 10, более 11, более 12, более 13, более 14, более 15, более 16, более 17 экзонов, более 18, более 19, более 20, более 21, более 22, более 23, более 24, более 25, более 26, более 27, более 28, более 29, более 30, более 31, более 32, более 33, более 34, более 35, более 36, более 37, более 38, более 39, более 40, более 41, более 42, более 43, более 44, более 45, более 46, более 47, более 48, более 49, более 50 экзонов с использованием одного единственного олигонуклеотида, и, таким образом, это пропускание более 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15,16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49 или 50 экзонов проводят, не используя смесь или коктейль двух или более различных олигонуклеотидов, или не используя два или более различных олигонуклеотидов, которые могут быть связаны с помощью одного или нескольких линкера(ов), или не используя генную конструкцию, транскрибирующую два или более различных олигонуклеотидов. В одном варианте осуществления, таким образом, предусматривается, что изобретение охватывает один единственный олигонуклеотид и не содержит двух или более различных олигонуклеотидов, причем указанный единственный олигонуклеотид способен связываться с указанным первым и указанным вторым экзонами и способен индуцировать пропускание по меньшей мере указанного первого и указанного второго экзонов в одной пре-мРНК, как проиллюстрировано в настоящем описании. В этом контексте квалифицированному специалисту будет понятно, что слово "единственный" не относится к количеству молекул, требуемому для индукции пропускания экзонов. "Единственный" относится к последовательности олигонуклеотида: изобретение охватывает одну единственную олигонуклеотидную последовательность и ее применение, и не включает две или более различных олигонуклеотидных последовательностей, причем указанная единственная олигонуклеотидная последовательность способна связываться с указанным первым и указанным вторым экзонами и способна индуцировать пропускание по меньшей мере указанного первого и указанного второго экзонов в одной пре-мРНК, как объяснено в настоящем описании. Это изобретение представляет собой первое изобретение, позволяющее пропускание более чем одного экзона с помощью только одного единственного олигонуклеотида для лечения заболевания, вызываемого (редкой) мутацией в гене, при условии, что различные экзоны в указанном гене включают области, которые обладают по меньшей мере 50% идентичностью последовательностей.
Олигонуклеотид по изобретению предпочтительно используют в качестве части терапии на основе активности модулирования РНК, как определено в настоящем описании ниже. В зависимости от типа транскрипта, где присутствуют первый и второй экзоны, можно сконструировать олигонуклеотид для предупреждения, лечения или замедления данного заболевания.
Было показано, что нацеливание на два экзона в одной пре-мРНК с помощью одного олигонуклеотида, способного связываться с обоими экзонами, приводит к мРНК, лишенной экзонов-мишеней, и, кроме того, всего участка экзонов между ними. Преимуществом такого единственного олигонуклеотида, как определено в настоящем описании, является то, что можно лечить дефекты, вызванные различными мутациями в этом мультиэкзонном участке. Если выбрать два или более различных олигонуклеотидов для индукции пропускания двух или более экзонов, следует, например, учитывать, что каждый олигонуклеотид может обладать его собственным профилем PK, и что, таким образом, необходимо будет найти условия, где каждый из них будет сходным образом присутствовать в одной и той же клетке. Таким образом, другим преимуществом использования только одного единственного олигонуклеотида, способного связываться с двумя различными экзонами, как определено выше, является то, что значительно упрощается изготовление, исследование токсичности, поиск дозы и клиническое испытание, поскольку это может быть сведено к простому изготовлению и исследованию одного единственного соединения.
Ниже определены дополнительные признаки олигонуклеотида по изобретению.
В контексте изобретения, в предпочтительном варианте осуществления олигонуклеотид способен связываться с, нацеливаться на, гибридизоваться с, является обратно комплементарным и/или способен ингибировать функцию по меньшей мере одной регулирующей сплайсинг последовательности в по меньшей мере указанном первом экзоне и/или указанном втором экзоне и/или влияет на структуру по меньшей мере указанного первого экзона и/или указанного второго экзона:
где указанный олигонуклеотид содержит последовательность, которая способна связываться с, нацеливаться на, гибридизоваться с и/или является обратно комплементарной участку связывания серин-аргининового (SR) белка в указанном первом и/или втором экзоне,
и/или
где указанный олигонуклеотид способен связываться с, нацеливаться на, гибридизоваться с и/или является обратно комплементарным энхансеру сплайсинга экзонов (ESE), последовательности распознавания экзонов (ERS) и/или сайленсеру сплайсинга экзонов (ESS) в указанном первом и/или втором экзоне.
Более предпочтительно, указанный олигонуклеотид, который способен связываться с, нацеливаться на, гибридизоваться с и/или является обратно комплементарным области первого экзона пре-мРНК и/или области второго экзона пре-мРНК, способен специфически ингибировать по меньшей мере одну регулирующую сплайсинг последовательность и/или влиять на структуру по меньшей мере указанного первого и/или второго экзона в указанной пре-мРНК. Препятствование регулирующим сплайсинг последовательностям и/или структурам имеет преимущество, состоящее в том, что такие элементы располагаются в экзоне. Путем предоставления такого олигонуклеотида, как определено в настоящем описании, можно эффективно замаскировать по меньшей мере указанный первый и второй экзоны, и предпочтительно полный участок экзонов между ними, от аппарата сплайсинга. Отсутствие распознавания аппаратом сплайсинга этих экзонов, таким образом, приводит к пропусканию или исключению этих экзонов из конечной мРНК. Этот вариант осуществления сфокусирован только на кодирующих последовательностях. Полагают, что это позволяет способу быть более специфическим и, таким образом, надежным. Обратная комплементарность указанного олигонуклеотида с указанной областью указанного первого и/или второго экзона в пре-мРНК предпочтительно составляет по меньшей мере 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% или 100%.
Таким образом, настоящее изобретение относится к антисмысловому олигонуклеотиду для применения в качестве лекарственного средства для предупреждения, замедления, смягчения и/или лечения заболевания у индивидуума,
где указанный олигонуклеотид способен связываться с областью первого экзона и областью второго экзона, где указанная область указанного второго экзона обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона,
где указанный первый и указанный второй экзоны находятся в одной и той же пре-мРНК у указанного индивидуума,
где указанное связывание приводит к пропусканию указанного первого экзона и указанного второго экзона и предпочтительно к пропусканию мультиэкзонного участка, начинающегося с указанного первого экзона и охватывающего один или несколько экзонов, находящихся между указанным первым и указанным вторым экзонами, и в лучшем случае к пропусканию всего участка экзонов между указанным первым и указанным вторым экзонами, и
где получают транскрипт в рамке считывания, позволяющий продуцирование функционального или полуфункционального белка.
Предпочтительно, как проиллюстрировано в настоящем описании, указанный олигонуклеотид способен индуцировать пропускание всего участка экзонов между указанным первым экзоном и указанным вторым экзоном.
Более предпочтительно, как объяснено в настоящем описании, указанное связывание указанного олигонуклеотида способно препятствовать по меньшей мере одной регулирующей сплайсинг последовательности в указанных областях указанных первого и второго экзонов, и/или вторичной структуре указанного первого и/или указанного второго экзонов, и/или вторичной структуре, охватывающей по меньшей мере указанный первый и/или указанный второй экзоны в указанной пре-мРНК. Предпочтительные регулирующие сплайсинг последовательности представлены в настоящем описании ниже.
Таким образом, один предпочтительный вариант осуществления относится к олигонуклеотиду по изобретению, который способен связываться с областью первого экзона в пре-мРНК и/или с областью второго экзона в той же пре-мРНК, где указанная область указанного второго экзона в той же пре-мРНК обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона (например, для DMD, предпочтительно, как в таблице 2), где указанный олигонуклеотид способен индуцировать пропускание указанного первого и указанного второго экзонов в указанной пре-мРНК; что приводит к транскрипту, который находится в рамке считывания (например, для DMD, предпочтительно, как в таблице в таблице 1 или 6). Указанный олигонуклеотид обеспечивает у указанного индивидуума функциональный или полуфункциональный белок, и указанный олигонуклеотид дополнительно содержит:
- последовательность, которая способна связываться с, нацеливаться на, гибридизоваться с и/или является обратно комплементарной области первого и/или второго экзона пре-мРНК, которая гибридизуется с другой частью первого и/или второго экзона пре-мРНК (закрытая структура), и/или
- последовательность, которая способна связываться с, нацеливаться на, гибридизоваться с и/или является обратно комплементарной области указанного первого и/или второго экзона пре-мРНК, которая не гибридизуется с указанной пре-мРНК (открытая структура).
Для этого варианта осуществления приводится ссылка на патентную заявку WO 2004/083446. Молекулы РНК обладают прочными вторичными структурами, по большей части вследствие спаривания оснований обратно комплементарных или частично обратно комплементарных участков в одной и той же РНК. Уже давно полагают, что структуры в РНК играют роль в функции РНК. Без связи с теорией, полагают, что вторичная структура РНК экзона играет роль в структурировании процесса сплайсинга. В ее структуре экзон распознается в качестве части, которая должна быть включена в мРНК. В одном варианте осуществления олигонуклеотид способен препятствовать структуре по меньшей мере указанного первого экзона и, возможно, также указанного второго экзона и, возможно, также участка экзонов между ними, и, таким образом, способен препятствовать сплайсингу указанного первого, и, возможно, также указанного второго экзона, и, возможно, также участка экзонов между ними, путем маскирования указанных экзонов от аппарата сплайсинга и тем самым обеспечения пропускания указанных экзонов. Без связи с теорией, полагают, что перекрывание с открытой структурой повышает эффективность инвазии олигонуклеотида (т.е. повышает эффективность, с которой олигонуклеотид может проникать в структуру), в то время как перекрывание с закрытой структурой повышает эффективность препятствования вторичной структуре РНК экзона. Было обнаружено, что длина частичной обратной комплементарности как с закрытой, так и с открытой структурой в значительной степени не ограничена. Авторы настоящего изобретения наблюдали высокую эффективность в случае олигонуклеотида с различной длиной обратной комплементарности в любой структуре. Термин "обратная комплементарность" используют в настоящем описании для обозначения участка нуклеиновых кислот, которые могут гибридизоваться с другим участком нуклеиновых кислот в физиологических условиях. Условия гибридизации определены в настоящем описании ниже. Таким образом, не является абсолютным требованием, чтобы все основания в области обратной комплементарности были способны спариваться с основаниями на противоположной цепи. Например, при конструировании олигонуклеотида может быть желательным включение, например, одного или нескольких остатков, которые не образуют пары оснований с основаниями на обратной комплементарной цепи. Несоответствия оснований могут быть до некоторой степени допустимыми, если в условиях в клетке участок нуклеотидов все еще способен гибридизоваться с обратно комплементарной частью. В контексте настоящего изобретения присутствие несоответствия в олигонуклеотиде по изобретению является предпочтительным, поскольку в одном варианте осуществления указанный олигонуклеотид является по меньшей мере на 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 100% обратно комплементарным области первого экзона и/или области второго экзона. Присутствие несоответствия в указанном олигонуклеотиде является предпочтительной характеристикой изобретения, поскольку указанный олигонуклеотид способен связываться с областью указанного первого и с областью указанного второго экзона, как определено в настоящем описании выше.
Другие преимущества обеспечения присутствия несоответствия оснований в антисмысловом олигонуклеотиде по изобретению определены в настоящем описании, и они сходны с преимуществами, обеспечиваемыми присутствием инозина (гипоксантина), и/или универсального основания и/или вырожденного основания и/или нуклеотида, содержащего основание, способное образовывать качающуюся пару оснований: избегание присутствия CpG, избегание или снижение потенциальной мультимеризации или агрегации, избегание квадруплексных структур, возможность конструирования олигонуклеотида с улучшенной кинетикой связывания РНК и/или термодинамическими свойствами.
Предпочтительно, обратная комплементарность олигонуклеотида области идентичности между указанными первым и/или вторым экзонами составляет от 45% до 65%, от 50% до 75%, но более предпочтительно от 65% до 100% или от 70% до 90%, или от 75% до 85%, или от 80% до 95%. Как правило, это обеспечивает 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или 11 несоответствий в олигонуклеотиде из 20 нуклеотидов. Таким образом, в олигонуклеотиде из 40 нуклеотидов может быть 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 18, 19, 20, 21 или 22 несоответствия оснований. Предпочтительно, в олигонуклеотиде из 40 нуклеотидов присутствует менее 14 несоответствий оснований. Количество несоответствий оснований является таким, что олигонуклеотид по изобретению все еще способен связываться с, гибридизоваться с, нацеливаться на область указанного первого экзона и на область указанного второго экзона, тем самым индуцируя пропускание по меньшей мере указанного первого и указанного второго экзонов и индуцируя продукцию транскрипта в рамке считывания, как объяснено в настоящем описании. Предпочтительно, продукции транскрипта в рамке считывания достигают по меньшей мере с 5% эффективностью при использовании по меньшей мере 100 нМ указанного олигонуклеотида для трансфекции соответствующей клеточной культуры in vitro, как объяснено в настоящем описании выше.
Следует отметить, что изобретение охватывает олигонуклеотид, который не обладает никаким несоответствием оснований с областью первого экзона и который может иметь 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 18, 19, 20, 21 или 22 несоответствия оснований с соответствующей областью второго экзона, как определено в настоящем описании. Однако изобретение также охватывает олигонуклеотид, который не имеет никакого несоответствия оснований с областью второго экзона и который может иметь 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 18, 19, 20, 21 или 22 несоответствия оснований с соответствующей областью первого экзона, как определено в настоящем описании. Наконец, изобретение охватывает олигонуклеотид, который может иметь 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 18, 19, 20, 21 или 22 несоответствия оснований с областью первого экзона и который может иметь 22, 21, 20, 19, 18, 17, 16, 15, 14, 13, 12, 11, 10, 9, 8, 7, 6, 5, 4, 3, 2, 1 несоответствие оснований с соответствующей областью второго экзона, как определено в настоящем описании. Вновь, в рамках настоящего изобретения следует понимать, что количество несоответствий оснований в олигонуклеотиде по изобретению является таким, чтобы этот указанный олигонуклеотид все еще был способен связываться с, гибридизоваться с, нацеливаться на область указанного первого экзона и область указанного второго экзона, как пояснено в настоящем описании.
Олигонуклеотид по изобретению предпочтительно не пересекает пропуск в выравнивании области первого экзона и области второго экзона, как определено в настоящем описании. Выравнивание предпочтительно проводят с использованием сетевого инструмента EMBOSS Matcher, как пояснено в настоящем описании выше. Однако в конкретных случаях возможно, что олигонуклеотид по изобретению должен пересекать пропуск, как упомянуто в настоящем описании выше. Предпочтительно, указанный пропуск представляет собой только один пропуск. Указанный пропуск предпочтительно охватывает менее 3 нуклеотидов, наиболее предпочтительно только один нуклеотид. Количество и длина пропусков являются такими, что олигонуклеотид по изобретению все еще способен связываться с, гибридизоваться с, нацеливаться на область указанного первого экзона и область указанного второго экзона, тем самым индуцируя пропускание по меньшей мере указанного первого и указанного второго экзонов и индуцируя продукцию транскрипта в рамке считывания, как пояснено в настоящем описании.
Структуру (т.е. открытые и закрытые структуры) наилучшим образом анализируют в контексте пре-мРНК, где находится экзон. Такую структуру можно анализировать в истинной РНК. Однако в настоящее время возможно спрогнозировать вторичную структуру молекулы РНК (с наибольшими затратами энергии) достаточно тщательно с использованием моделирующих структуру программ. Неограничивающим примером подходящей программы является интернет-сервер Mfold (Zuker, M.).
Специалист в данной области способен спрогнозировать с приемлемой воспроизводимостью вероятную структуру экзона, учитывая нуклеотидную последовательность. Наилучшее прогнозирование осуществляют, когда программе предоставляют как последовательность указанного экзона, так и последовательности фланкирующих интронов. Как правило, не является необходимым моделирование структуры всей пре-мРНК.
Отжиг олигонуклеотида по изобретению может влиять на локальное сворачивание или 3D-структуру или конформацию РНК-мишени (т.е. область, охватывающую по меньшей мере первый и/или второй экзоны). Отличающаяся конформация может приводить к нарушению структуры, распознаваемой аппаратом сплайсинга. Однако, когда в первом и/или втором экзоне-мишени присутствуют потенциальные (скрытые) акцепторные и/или донорные последовательности сплайсинга, иногда образуется новая структура, определяющая отличающийся (нео-) экзон, т.е. с отличающимся 5'-концом, отличающимся 3'-концом или обоими из них. Этот тип активности находится в объеме настоящего изобретения, поскольку экзон-мишень исключается из мРНК. Присутствие нового экзона, содержащего часть указанного первого и/или второго экзона-мишени в мРНК, не изменяет тот факт, что экзон-мишень, как таковой, исключается. Включение нового экзона можно наблюдать в качестве побочного эффекта, который происходит только изредка. Существует две возможности, когда пропускание экзона используется для восстановления (части) открытой рамки считывания транскрипта, которая разрушается в результате мутации. Одной из них является то, что новый экзон является функциональным в отношении восстановления рамки считывания, в то время как в другом случае рамка считывания не восстанавливается. При выборе олигонуклеотида для восстановления открытой рамки считывания с помощью пропускания множества экзонов, безусловно, очевидно, что в этих условиях выбирают только те олигонуклеотиды, которые действительно приводят к пропусканию экзона, которое восстанавливает открытую рамку считывания данного транскрипта, с новым экзоном или без него.
Кроме того, в другом предпочтительном варианте осуществления предусматривается олигонуклеотид по изобретению, который способен связываться с областью первого экзона в пре-мРНК и с областью второго экзона в той же пре-мРНК, где указанная область указанного второго экзона обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона (предпочтительные области для экзонов дистрофина указаны в таблице 2 или таблице 6), где указанный олигонуклеотид способен индуцировать пропускание указанных первого и второго экзонов указанной пре-мРНК; что приводит к транскрипту, который находится в рамке считывания (для DMD, предпочтительно, как в таблице 1 или таблице 6). Указанный олигонуклеотид обеспечивает у указанного индивидуума функциональный или полуфункциональный белок, и указанный олигонуклеотид дополнительно содержит: последовательность, которая способна связываться с, нацеливаться на, гибридизоваться с, является обратно комплементарной для и/или способна ингибировать функцию одного или нескольких участков связывания серин-аргининового (SR) белка в РНК экзона из пре-мРНК.
В патентной заявке WO 2006/112705 авторы настоящего изобретения описали наличие корреляции между эффективностью внутреннего антисмыслового олигонуклеотида (AON) экзона в отношении индукции пропускания экзонов и присутствием предполагаемого участка связывания SR в заданном участке пре-мРНК указанного AON. Таким образом, в одном варианте осуществления получают указанный олигонуклеотид, как определено в настоящем описании, включая определение одного или нескольких (предполагаемых) участков связывания для белка SR в РНК указанного первого и/или указанного второго экзона и получение соответствующего олигонуклеотида, который способен связываться с, нацеливаться на, гибридизоваться с и/или является обратно комплементарным указанной РНК и который по меньшей мере частично перекрывает указанный (предполагаемый) участок связывания. Термин "по меньшей мере частично перекрывает" определяют в настоящем описании как включающий перекрывание только одного нуклеотида участка связывания SR, а также множества нуклеотидов одного или нескольких указанных участков связывания, а также полное перекрывание одного или нескольких указанных участков связывания. Этот вариант осуществления, предпочтительно, дополнительно включает определение во вторичной структуре первого и/или второго экзона области, которая гибридизуется с другой частью указанного первого и/или второго экзона (закрытая структура), и области, которая не гибридизуется в указанной структуре (открытая структура), а затем получение олигонуклеотида, который по меньшей мере частично перекрывает один или несколько указанных (предполагаемых) участков связывания, и который перекрывает по меньшей мере часть указанной закрытой структуры и перекрывает по меньшей мере часть указанной открытой структуры, и который связывается с, нацеливается на, гибридизуется с и/или является обратно комплементарным первому и второму экзонам. Таким образом, авторы настоящего изобретения увеличивают вероятность получения олигонуклеотида, который способен препятствовать включению указанного первого и второго экзонов, и, если это применимо, полного участка экзонов между ними, из пре-мРНК в мРНК. Без связи с какой-либо теорией, в настоящее время полагают, что применение олигонуклеотида, направленного на участок связывания белка SR, приводит (по меньшей мере частично) к нарушению связывания белка SR с указанным участком связывания, что приводит к нарушенному или ухудшенному сплайсингу.
Предпочтительно, область первого экзона и/или область второго экзона в одной и той же пре-мРНК, с которой способен связываться олигонуклеотид по изобретению, содержит открытую/закрытую структуру и/или участок связывания белка SR, более предпочтительно указанная открытая/закрытая структура и указанный участок связывания белка SR частично перекрываются, и еще более предпочтительно указанная открытая/закрытая структура полностью перекрывает участок связывания белка SR, или участок связывания белка SR полностью перекрывает открытую/закрытую структуру. Это обеспечивает дальнейшее нарушение включения экзона.
Помимо консенсусных последовательностей участков связывания, множество (или даже все) экзонов содержат регулирующие сплайсинг последовательности, такие как последовательности энхансера сплайсинга экзонов (ESE), чтобы облегчить распознавание истинных участков сплайсинга сплайсингосомой (Cartegni L, et al. 2002; и Cartegni L, et al., 2003). Подгруппа факторов связывания, называемых белками SR, может связываться с этими ESE и привлекать другие факторы сплайсинга, такие как U1 и U2AF, в (слабо определенные) участки сплайсинга. Участки сплайсинга четырех из наиболее распространенных белков SR (SF2/ASF, SC35, SRp40 и SRp55) проанализированы детально (Cartegni L, et al. 2002 и Cartegni L, et al., 2003). Существует корреляция между эффективностью олигонуклеотида и наличием/отсутствием участка связывания SF2/ASF, SC35, SRp40 и SRp55 в части первого экзона, с которой связывается, гибридизуется и/или на которую нацеливается указанный олигонуклеотид. В предпочтительном варианте осуществления изобретение, таким образом, относится к олигонуклеотиду, который связывается с, гибридизуется с, нацеливается на и/или обратно комплементарен участку связывания для белка SR. Предпочтительно, указанный SR белок представляет собой SF2/ASF или SC35, SRp40 или SRp55. В одном варианте осуществления олигонуклеотид связывается с, гибридизуется с, нацеливается на и/или является обратно комплементарным участку связывания белка SF2/ASF, SC35, SRp40 или SRp55 в первом экзоне и участку связывания другого белка SF2/ASF, SC35, SRp40 или SRp55 во втором экзоне. В более предпочтительном варианте осуществления олигонуклеотид связывается с, гибридизуется с, нацеливается на и/или является обратно комплементарным участку связывания белка SF2/ASF, SC35, SRp40 или SRp55 в первом экзоне и участку связывания сходного белка SF2/ASF, SC35, SRp40 или SRp55 во втором экзоне.
В одном варианте осуществления у пациента обеспечивают функциональный или полуфункциональный белок с использованием олигонуклеотида, который способен связываться с, нацеливаться на регуляторную последовательность РНК, присутствующую в первом и/или втором экзоне, которая требуется для правильного сплайсинга указанного экзона(ов) в транскрипте. Для правильного сплайсинга экзонов в транскрипте требуется несколько цис-действующих последовательностей РНК. В частности, идентифицированы дополнительные элементы, такие как энхансеры сплайсинга экзонов (ESE), для регуляции специфического и эффективного сплайсинга конститутивных и альтернативных экзонов. С использованием соединения, содержащего олигонуклеотид, который связывается или способен связываться с одним из дополнительных элементов в указанном первом и/или втором экзоне(ах), их регуляторная функция нарушается так, что экзоны пропускаются. Таким образом, в одном предпочтительном варианте осуществления олигонуклеотид по изобретению способен связываться с областью первого экзона в пре-мРНК и областью второго экзона в той же пре-мРНК, где указанная область указанного второго экзона обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона, где указанный олигонуклеотид способен индуцировать пропускание указанных первого и второго экзона в указанной пре-мРНК; где указанная область указанного первого экзона и/или указанная область указанного второго экзона содержит энхансер сплайсинга экзонов (ESE), последовательность распознавания экзонов (ERS) и/или последовательность энхансера сплайсинга (SES), и/или где указанный олигонуклеотид способен связываться с, нацеливаться на, способен ингибировать и/или является обратно комплементарным указанному энхансеру сплайсинга экзонов (ESE), последовательности распознавания экзонов (ERS) и/или последовательности энхансера сплайсинга (SES).
Ниже описаны предпочтительные химические структуры олигонуклеотида по изобретению.
Олигонуклеотид широко известен как олигомер, который имеет базовые характеристики гибридизации, сходные с природными нуклеиновыми кислотами. Гибридизация определена в разделе, посвященном определениям, в конце описания изобретения. В настоящей заявке термины "олигонуклеотид" и "олигомер" используют взаимозаменяемо. Для получения указанного олигонуклеотида по изобретению можно использовать различные типы нуклеозидов. Олигонуклеотид может содержать по меньшей мере одну модифицированную межнуклеозидную связь и/или по меньшей мере одну модификацию сахара и/или по меньшей мере одну модификацию основания по сравнению с встречающимся в природе олигонуклеотидом на основе рибонуклеотида или дезоксирибонуклеотида.
"Модифицированная межнуклеозидная связь" указывает на присутствие модифицированной версии фосфодиэфира относительно встречающейся в природе РНК и ДНК. Примерами модификаций межнуклеозидной связи, которые совместимы с настоящим изобретением, являются фосфоротиоат (PS), хирально чистый фосфоротиоат, фосфородитиоат (PS2), фосфоноацетат (PACE), фосфоноацетамид (PACA), тиофосфоноацетат, тиофосфоноацетамид, фосфоротиоатное пролекарство, H-фосфонат, метилфосфонат, метилфосфонотиоат, метилфосфат, метилфосфоротиоат, этилфосфат, этилфосфоротиоат, боранофосфат, боранофосфоротиоат, метилборанофосфат, метилборанофосфоротиоат, метилборанофосфонат, метилборанофосфонотиоат и их производные. Другая модификация включает фосфорамидит, фосфорамидат, N3'→P5' фосфорамидат, фосфородиамидат, фосфортиоамидат, фосфоротиодиамидат, сульфамат, диметиленсульфоксид, сульфонат, метиленимино (MMI), оксалил- и тиоацетамидонуклеиновую кислоту (TANA); и их производные. В зависимости от длины, олигонуклеотид по изобретению может содержать 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38 или 39 модификаций основной цепи. Также изобретение охватывает внесение в указанный олигонуклеотид более одной отличающейся модификации основной цепи.
Также изобретение охватывает олигонуклеотид, который содержит межнуклеозидную связь, которая может отличаться с точки зрения атомов нуклеозидов, которые связаны друг с другом, по сравнению со встречающейся в природе межнуклеозидной связью. В этом отношении, олигонуклеотид по изобретению может содержать по меньшей мере одну межнуклеозидную связь, сконструированную как мономеры, связанные 3'-3', 5'-5', 2'-3', 2'-5', 2'-2'. Нумерация положений может отличаться для других химических структур, однако идея сохраняется в объеме изобретения.
В одном варианте осуществления олигонуклеотид по изобретению содержит по меньшей мере одну фосфоротиоатную модификацию. В более предпочтительном варианте осуществления олигонуклеотид по изобретению является полностью модифицированным посредством фосфоротиоата.
"Модификация сахара" указывает на присутствие модифицированной версии рибозильной части относительно рибозильной части во встречающихся в природе РНК и ДНК (т.е. фуранозильная часть), такой как бициклические сахара, тетрагидропираны, структуры морфолино, 2'-модифицированные сахара, 3'-модифицированные сахара, 4'-модифицированные сахара, 5'-модифицированные сахара и 4'-замещенные сахара. Примеры подходящих модификаций сахаров включают, но не ограничиваются ими, 2'-O-модифицированные нуклеотидные остатки РНК, такие как 2'-O-алкил или 2'-O-(замещенный)алкил, например 2'-O-метил, 2'-O-(2-цианоэтил), 2'-O-(2-метокси)этил (2'-MOE), 2'-O-(2-тиометил)этил, 2'-O-бутирил, 2'-O-пропаргил, 2'-O-аллил, 2'-O-(2-амино)пропил, 2'-O-(2-(диметиламино)пропил), 2'-O-(3-амино)пропил, 2'-O-(3-(диметиламино)пропил), 2'-O-(2-амино)этил, 2'-O-(3-гуанидино)пропил (как описано в патентной заявке WO 2013/061295, University of the Witwatersrand, включенной в настоящее описание в качестве ссылки), 2'-O-(2-(диметиламино)этил); 2'-O-(галогеналкокси)метил (Arai K. et al.), например, 2'-O-(2-хлорэтокси)метил (MCEM), 2'-O-(2,2-дихлорэтокси)метил (DCEM); 2'-O-алкоксикарбонил, например, 2'-O-[2-(метоксикарбонил)этил] (MOCE), 2'-O-[2-(N-метилкарбамоил)этил] (MCE), 2'-O-[2-(N,N-диметилкарбамоил)этил] (DMCE); 2'-O-[метиламинокарбонил]метил; 2'-азидо; 2'-амино и 2'-замещенный амино; 2'-галоген, например, 2'-F, FANA (2'-F арабинозилнуклеиновая кислота); карбо- и аза- модификации сахаров; 3'-O-алкил, например, 3'-O-метил, 3'-O-бутирил, 3'-O-пропаргил; 2',3'-дидезокси; и их производные.
Другая модификация сахара включает "мостиковые" или "бициклические" (BNA) модифицированные части сахаров нуклеиновой кислоты, такие, как встречаются, например, в закрытой нуклеиновой кислоте (LNA), ксило-LNA, α-L-LNA, β-D-LNA, cEt (2'-O,4'-C связанный этил) LNA, cMOEt (2'-O,4'-C связанный метоксиэтил) LNA, связанной этиленовым мостиком нуклеиновой кислоте (ENA), BNANC[N-Me] (как описано в Chem. Commun. 2007, 3765-3767 Kazuyuki Miyashita et al., которая включена в настоящее описание в качестве ссылки в полном объеме), CRN, как описано в патентной заявке WO 2013/036868 (Marina Biotech, включенной в настоящее описание в качестве ссылки в полном объеме); разомкнутой нуклеиновой кислоте (UNA) или других ациклических нуклеозидах, таких как описаны в патентной заявке США US 2013/0130378 (Alnylam Pharmaceuticals), включенной в настоящее описание в качестве ссылки в полном объеме; 5'-метил-замещенных BNA (как описано в патентной заявке США 13/530218, которая включена в качестве ссылки в полном объеме); циклогексенилнуклеиновой кислоте (CeNA), алтриолнуклеиновой кислоте (ANA), гекситолнуклеиновой кислоте (HNA), фторированной HNA (F-HNA), пиранозил-РНК (p-РНК), 3'-дезоксипиранозил-ДНК (p-ДНК); или других модифицированных частях сахаров, таких как морфолино (PMO), катионный морфолино (PMOPlus), PMO-X; трициклоДНК; трицикло-PS-ДНК; и их производные. Производные BNA описаны, например, в WO 2011/097641, которая включена в настоящее описание в качестве ссылки в полном объеме. Примеры PMO-X описаны в WO 2011150408, которая включена в настоящее описание в качестве ссылки в полном объеме. В зависимости от длины, олигонуклеотид по изобретению может содержать 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39 или 40 модификаций сахаров. Также изобретение охватывает внесение более одной отличающейся модификации сахаров в указанный олигонуклеотид.
В одном варианте осуществления олигонуклеотид по изобретению содержит по меньшей мере одну модификацию сахара, выбранную из 2'-O-метила, 2'-O-(2-метокси)этила, 2'-F, морфолино, мостикового нуклеотида или BNA, или олигонуклеотид содержит как мостиковые нуклеотиды, так и 2'-дезоксинуклеотиды (смешанные BNA/ДНК-меры). Было показано, что олигонуклеотиды, содержащие 2'-фтор (2-'F) нуклеотид, способны привлекать связывающий энхансер интерлейкина фактор 2 и 3 (ILF2/3) и тем самым способны индуцировать пропускание экзонов в пре-мРНК-мишени (Rigo F, et al., WO 2011/097614).
В другом варианте осуществления олигонуклеотид, как определено в настоящем описании, содержит или состоит из LNA или ее производного. Более предпочтительно, олигонуклеотид по изобретению модифицирован по его всей длине модификацией сахара, выбранной из 2'-O-метила, 2'-O-(2-метокси)этила, морфолино, мостиковой нуклеиновой кислоты (BNA) или смешанного BNA/ДНК-мера. В более предпочтительном варианте осуществления олигонуклеотид по изобретению является полностью 2'-O-метил-модифицированным.
В предпочтительном варианте осуществления олигонуклеотид по изобретению содержит по меньшей мере одну модификацию сахара и по меньшей мере одну модифицированную межнуклеозидную связь. Такие модификации включают пептидно-нуклеиновую кислоту (PNA), модифицированную борным кластером PNA, окси-пептидно-нуклеиновую кислоту на основе пирролидина (POPNA), нуклеиновую кислоту на основе гликоля или глицерина (GNA), нуклеиновую кислоту на основе треозы (TNA), ациклическую нуклеиновую кислоту на основе треонинола (aTNA), олигонуклеотиды на основе морфолино (PMO, PPMO, PMO-X), катионные олигомеры на основе морфолино (PMOPlus, PMO-X), олигонуклеотиды с объединенными основаниями и остовами (ONIB), пирролидин-амидные олигонуклеотиды (POM); и их производные. В предпочтительном варианте осуществления олигонуклеотид по изобретению содержит остов пептидно-нуклеиновой кислоты и/или остов морфолинофосфородиамидата или их производное. В более предпочтительном варианте осуществления олигонуклеотид по изобретению является 2'-O-метилфосфоротиоат-модифицированным, т.е. содержит по меньшей мере одну 2'-O-метилфосфоротиоатную модификацию, предпочтительно олигонуклеотид по изобретению является полностью 2'-O-метилфосфоротиоат-модифицированным. Предпочтительно, 2'-O-метилфосфоротиоат-модифицированный олигонуклеотид или полностью 2'-O-метилфосфоротиоат-модифицированный олигонуклеотид представляет собой РНК-олигонуклеотид.
Термин "модификация основания" или "модифицированное основание", как определено в настоящем описании, относится к модификации встречающегося в природе основания в РНК и/или ДНК (т.е. пиримидинового или пуринового основания) или к синтезированным de novo основаниям. Такое синтезированное de novo основание может рассматриваться как "модифицированное" по сравнению с существующим основанием.
В дополнение к модификациям, описанным выше, олигонуклеотид по изобретению может содержать дополнительные модификации, такие как различные типы нуклеотидных остатков нуклеиновой кислоты или нуклеотидов, как описано ниже. Для получения олигонуклеотида по изобретению можно использовать различные типы нуклеотидных остатков нуклеиновой кислоты. Указанный олигонуклеотид может иметь по меньшей мере одну модификацию основной цепи и/или сахара и/или по меньшей мере одну модификацию основания по сравнению с олигонуклеотидом на основе РНК или ДНК.
Олигонуклеотид может содержать природные основания пурины (аденин, гуанин) или пиримидины (цитозин, тимин, урацил) и/или модифицированные основания, как определено ниже. В контексте изобретения урацил может быть заменен тимином.
Модификация оснований включает модифицированную версию природных пуриновых и пиримидиновых оснований (например, аденин, урацил, гуанин, цитозин и тимин), таких как гипоксантин, оротовая кислота, агматидин, лизидин, псевдоурацил, псевдотимин, N1-метилпсевдоурацил, 2-тиопиримидин (например, 2-тиоурацил, 2-тиотимин), 2,6-диаминопурин, G-зажим и его производные, 5-замещенный пиримидин (например, 5-галогенурацил, 5-метилурацил, 5-метилцитозин, 5-пропинилурацил, 5-пропинилцитозин, 5-аминометилурацил, 5-гидроксиметилурацил, 5-аминометилцитозин, 5-гидроксиметилцитозин, Super T), 5-октилпиримидин, 5-тиофенпиримидин, 5-октин-1-илпиримидин, 5-этинилпиримидин, 5-(пиридиламид), 5-изобутил, 5-фенил, как описано в патентной заявке US 2013/0131141 (RXi), включенной в настоящее описание в качестве ссылки в полном объеме; 7-деазагуанин, 7-деазааденин, 7-аза-2,6-диаминопурин, 8-аза-7-деазагуанин, 8-аза-7-деазааденин, 8-аза-7-деаза-2,6-диаминопурин, Super G, Super A и N4-этилцитозин, или их производные; N2-циклопентилгуанин (cPent-G), N2-циклопентил-2-аминопурин (cPent-AP) и N2-пропил-2-аминопурин (Pr-AP), или их производные; и вырожденные или универсальные основания, такие как 2,6-дифтортолуол, или отсутствующие основания, такие как абазические участки (например, 1-дезоксирибоза, 1,2-дидезоксирибоза, 1-дезокси-2-O-метилрибоза; или производные пирролидина, в которых кислород кольца заменен азотом (азарибоза)). Примеры производных Super A, Super G и Super T могут быть найдены в патенте США 6683173 (Epoch Biosciences), который включен в настоящее описание в качестве ссылки. Было показано, что cPent-G, cPent-AP и Pr-AP снижает иммуностимулирующие эффекты при включении в миРНК (siRNA) (Peacock H. et al.), и сходные признаки были показаны для псевдоурацила и N1-метилпсевдоурацила (патентная заявка США 2013/0123481, modeRNA Therapeutics, включенная в настоящее описание качестве ссылки в полном объеме).
"Тимин" и "5-метилурацил" могут использоваться в настоящем документе взаимозаменяемо. Аналогично, "2,6-диаминопурин" идентичен "2-аминоаденину", и эти термины могут использоваться в настоящем документе взаимозаменяемо.
В предпочтительном варианте осуществления олигонуклеотид по изобретению содержит по меньшей мере один 5-метилцитозин и/или по меньшей мере один 5-метилурацил и/или по меньшей мере одно 2,6-диаминопуриновое основание, что следует понимать как то, что по меньшей мере одно из цитозиновых нуклеиновых оснований указанного олигонуклеотида модифицировано путем замены протона в положении 5 кольца пиримидина метильной группой (т.е. 5-метилцитозином), и/или что по меньшей мере одно из урацильных нуклеиновых оснований указанного олигонуклеотида модифицировано путем замены протона в положении 5 кольца пиримидина метильной группой (т.е. 5-метилурацил), и/или что по меньшей мере одно из адениновых нуклеиновых оснований указанного олигонуклеотида модифицировано путем замены протона в положении 2 аминогруппой (т.е. 2,6-диаминопурин), соответственно. В контексте изобретения выражение "замена протона метильной группой в положении 5 кольца пиримидина" может быть заменено выражением "замена пиримидина 5-метилпиримидином", причем пиримидин обозначает только урацил, только цитозин или оба из них. Аналогично, в контексте изобретения выражение "замена протона аминогруппой в положении 2 аденина" может быть заменено выражением "замена аденина 2,6-диаминопурином". Если указанный олигонуклеотид содержит 1, 2, 3, 4, 5, 6, 7, 8, 9 или более остатков цитозина, урацила и/или аденина, по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9 или более остатков цитозина, урацила и/или аденина, соответственно, могут быть модифицированы таким путем. В предпочтительном варианте осуществления все остатки цитозина, все остатки урацила и/или все остатки аденина модифицированы таким путем или заменены 5-метилцитозином, 5-метилурацилом и/или 2,6-диаминопурином, соответственно.
Было обнаружено, что присутствие 5-метилцитозина, 5-метилурацила и/или 2,6-диаминопурина в олигонуклеотиде по изобретению имеет положительный эффект по меньшей мере на один из параметров или улучшение по меньшей мере одного из параметров указанного олигонуклеотида. В этом контексте параметры могут включать: аффинность и/или кинетику связывания, активность сайленсинга, биостабильность, (внутритканевое) распределение, клеточный захват и/или транспорт, и/или иммуногенность указанного олигонуклеотида, как объяснено ниже.
Поскольку известно, что несколько модификаций, упомянутых выше, увеличивает величину Tm и, таким образом, усиливает связывание определенного нуклеотида с его аналогом на его мРНК-мишени, эти модификации могут быть исследованы в отношении усиления связывания олигонуклеотида по изобретению как с областью первого экзона, так и с областью второго экзона, в контексте изобретения. Поскольку последовательности олигонуклеотида по изобретению могут не быть на 100% обратно комплементарными данной области первого экзона и/или второго экзона, увеличивающие Tm модификации, такие как мостиковый нуклеотид или BNA (такой как LNA) или модификация основания, выбранная из 5-метилпиримидинов и/или 2,6-диаминопурина, предпочтительно могут быть осуществлены в нуклеотидном положении, которое обратно комплементарно указанной первой и/или указанной второй области указанных экзонов.
Аффинность связывания и/или кинетика связывания или гибридизации зависят от термодинамических свойств AON. Они по меньшей мере частично определяются температурой плавления указанного олигонуклеотида (Tm; вычисляемая, например, с помощью калькулятора свойств олигонуклеотидов (http://www.unc.edu/~cail/biotool/oligo/index.html или http://eu.idtdna.com/analyzer/Applications/OligoAnalyzer/) для одноцепочечной РНК с использованием базовой Tm и модели ближайшего соседа) и/или свободной энергией комплекса олигонуклеотида и экзона-мишени (с использованием RNA structure версии 4.5 или РНК mfold версии 3.5). Если Tm увеличивается, активность пропускания экзона, как правило, возрастает, однако ожидается, что, когда Tm является слишком высокой, AON станет менее специфичным к последовательности. Приемлемая Tm и свободная энергия зависят от последовательности олигонуклеотида. Таким образом, трудно привести предпочтительные диапазоны для каждого из этих параметров.
Активность олигонуклеотида по изобретению предпочтительно определяют следующим образом:
- смягчение одного или нескольких симптома(ов) заболевания, ассоциированного с мутацией, присутствующей в первом и/или во втором экзоне, и/или с мутацией, присутствующей в участке, начинающемся в указанном первом экзоне и оканчивающемся в указанном втором экзоне, предпочтительно смягчение одного или нескольких симптома(ов) DMD или BMD; и/или
- смягчение одной или нескольких характеристик клетки от пациента, предпочтительно мышечной клетки от пациента; и/или
- обеспечение у указанного индивидуума функционального или полуфункционального белка, предпочтительно функционального или полуфункционального белка дистрофина; и/или
- по меньшей мере частичное снижение продукции аберрантного белка у указанного индивидуума, предпочтительно по меньшей мере частичное снижение продукции аберрантного белка дистрофина у указанного индивидуума. Каждый из этих признаков и анализов для их оценки определен в настоящем описании ниже.
Ожидается, что предпочтительный олигонуклеотид по изобретению, содержащий 5-метилцитозин и/или 5-метилурацил и/или 2,6-диаминопуриновое основание, будет проявлять увеличенную активность по сравнению с соответствующей активностью олигонуклеотида без какого-либо 5-метилцитозина, без какого-либо 5-метилурацила и без какого-либо 2,6-диаминопуринового основания. Это различие в активности может составлять по меньшей мере 1%, 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% или 100%. Биораспределение и биостабильность предпочтительно по меньшей мере частично определяются валидированным анализом лигирования при гибридизации, взятого из Yu et al., 2002. В одном варианте осуществления образцы плазмы или образцы гомогенизированной ткани инкубируют с конкретным улавливающим олигонуклеотидным зондом. После разделения, DIG-меченный олигонуклеотид лигируют с комплексом и проводят детекцию с использованием связанной с антителом против DIG пероксидазой. Некомпартментный анализ фармакокинетики проводят с использованием пакета программ WINNONLIN (модель 200, версия 5.2, Pharsight, Mountainview, CA). Проводят мониторинг уровней AON (мкг) на мл плазмы или мг ткани с течением времени для оценки площади под кривой (AUC), максимальной концентрации (Cmax), времени до достижения максимальной концентрации (Tmax), терминального времени полужизни и латентного периода всасывания (tlag). Такой предпочтительный анализ описан в экспериментальной части.
Олигонуклеотид может стимулировать врожденный иммунный ответ путем активации Toll-подобных рецепторов (TLR), включая TLR9 и TLR7 (Krieg A.M., et al., 1995). Активация TLR9, как правило, происходит вследствие присутствия неметилированных последовательностей CG, присутствующих в олигодезоксинуклеотидах (ODN), посредством имитации бактериальной ДНК, которая активирует врожденную иммунную систему посредством опосредуемого TLR9 высвобождения цитокинов. Однако 2'-O-метил-модификация может значительно снижать такой возможный эффект. Описано, что TLR7 распознает урациловые повторы в РНК (Diebold S.S., et al., 2006).
Активация TLR9 и TLR7 приводит к набору скоординированных иммунных ответов, которые включают врожденный иммунитет (макрофаги, дендритные клетки (DC) и NK-клетки) (Krieg A.M., et al., 1995; Krieg A.M., et al. 2000). В этот процесс вовлечено несколько хемокинов и цитокинов, таких как IP-10, TNFα, IL-6, MCP-1 и IFNα (Wagner H., et al., 1999; Popovic P.J., et al., 2006). Воспалительные цитокины привлекают дополнительные защитные клетки из крови, такие как T- и B-клетки. Уровни этих цитокинов можно исследовать с помощью исследования in vitro. В кратком изложении, цельную кровь человека инкубируют с возрастающими концентрациями олигонуклеотидов, после чего уровни цитокинов определяют с помощью стандартных коммерчески доступных наборов для ELISA. Снижение иммуногенности предпочтительно соответствует поддающемуся обнаружению снижению концентрации по меньшей мере одного из цитокинов, упомянутых выше, при сравнении с концентрацией соответствующего цитокина в анализе в клетке, обработанной олигонуклеотидом, содержащим по меньшей мере один 5-метилцитозин и/или 5-метилурацил и/или 2,6-диаминопурин, по сравнению с клеткой, обработанной соответствующим олигонуклеотидом, не имеющим 5-метилцитозинов, 5-метилурацилов или 2,6-диаминопуринов.
Таким образом, предпочтительный олигонуклеотид по изобретению имеет улучшенный параметр, такой как приемлемая или сниженная иммуногенность и/или лучшее биораспределение и/или приемлемая или улучшенная кинетика связывания РНК и/или термодинамические свойства по сравнению с соответствующим олигонуклеотидом без 5-метилцитозина, без 5-метилурацила и без 2,6-диаминопурина. Каждый из этих параметров можно оценивать с использованием анализов, известных специалисту в данной области.
Предпочтительный олигонуклеотид по изобретению содержит или состоит из молекулы РНК или модифицированной молекулы РНК. В предпочтительном варианте осуществления олигонуклеотид является одноцепочечным. Однако квалифицированному специалисту будет понятно, что одноцепочечный олигонуклеотид может образовывать внутреннюю двухцепочечную структуру. Однако этот олигонуклеотид, тем не менее, называют одноцепочечным олигонуклеотидом в контексте настоящего изобретения. Одноцепочечный олигонуклеотид имеет несколько преимуществ по сравнению с двухцепочечным олигонуклеотидом миРНК: (i) ожидается, что его синтез будет более простым, чем синтез двух комплементарных цепей миРНК; (ii) существует более широкий диапазон химических модификаций, возможных для повышения захвата в клетки, более высокой (физиологической) стабильности и снижения потенциальных общих неблагоприятных эффектов; (iii) миРНК имеют более высокий потенциал для неспецифических эффектов (включая гены вне мишени) и улучшенную фармакологию (например, возможен меньший контроль эффективности и селективности схемы лечения или дозы); и (iv) миРНК с меньшей вероятностью будут действовать в ядре и не могут быть направлены против интронов.
В другом варианте осуществления олигонуклеотид по изобретению содержит абазический участок или абазический мономер. В контексте изобретения такой мономер может называться абазическим участком или абазическим мономером. Абазический мономер представляет собой нуклеотидный остаток или структурный элемент, который лишен нуклеинового основания, по сравнению с соответствующим нуклеотидным остатком, содержащим нуклеиновое основание. В рамках изобретения абазический мономер, таким образом, представляет собой часть структурного элемента олигонуклеотида, но лишенную нуклеинового основания. Такой абазический мономер может присутствовать, или может быть связан, или может быть присоединен, или может быть конъюгирован со свободным концом олигонуклеотида.
В более предпочтительном варианте осуществления олигонуклеотид по изобретению содержит 1-10 или более абазических мономеров. Таким образом, в олигонуклеотиде по изобретению может находиться 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более абазических мономеров.
Абазический мономер может представлять собой любой тип, известный и понятный квалифицированному специалисту, неограничивающие примеры которых представлены ниже:

В рамках настоящего изобретения, R1 и R2 независимо представляют собой H, олигонуклеотид или другой абазический участок(ки) при условии, что не оба из R1 и R2 представляют собой H и R1 и R2 не оба являются олигонуклеотидами. Абазический мономер(ы) может быть связан с любым или с обоими концами олигонуклеотида, как указано выше. Следует отметить, что олигонуклеотид, связанный с одним или двумя абазическим участком(ами) или абазическим мономером(ами), может содержать менее 10 нуклеотидов. В этом отношении олигонуклеотид по изобретению может содержать по меньшей мере 10 нуклеотидов, необязательно включающих один или несколько абазических участков или абазических мономеров на одном или обоих концах. Другие примеры абазических участков, которые охватываются изобретением, описаны в патентной заявке США 2013/013378 (Alnylam Pharmaceuticals), включенной в настоящее описание в качестве ссылки в полном объеме.
В зависимости от длины олигонуклеотид по изобретению может содержать 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39 или 40 модификаций оснований. Также изобретение охватывает внесение более одной определенной модификации оснований в указанный олигонуклеотид.
Таким образом, в одном варианте осуществления олигонуклеотид по изобретению содержит:
(a) по меньшей мере одну модификацию основания, выбранную из 2-тиоурацила, 2-тиотимина, 5-метилцитозина, 5-метилурацила, тимина, 2,6-диаминопурина; и/или
(b) по меньшей мере одну модификацию сахара, выбранную из 2'-O-метила, 2'-O-(2-метокси)этила, 2'-O-дезокси (ДНК), 2'-F, морфолино, мостикового нуклеотида или BNA, или олигонуклеотид содержит как мостиковые нуклеотиды, так и 2'-дезокси-модифицированные нуклеотиды (смешанные BNA/ДНК-меры); и/или
(c) по меньшей мере одну модификацию основной цепи, выбранную из фосфоротиоата или фосфородиамидата.
В другом варианте осуществления олигонуклеотид по изобретению содержит:
(a) по меньшей мере одну модификацию оснований, выбранную из 5-метилпиримидина и 2,6-диаминопурина; и/или
(b) по меньшей мере одну модификацию сахара, которая представляет собой 2'-O-метил; и/или
(c) по меньшей мере одну модификацию основной цепи, которая представляет собой фосфоротиоат.
В одном варианте осуществления олигонуклеотид по изобретению содержит по меньшей мере одну модификацию по сравнению со встречающимся в природе олигонуклеотидом на основе рибонуклеотида или дезоксирибонуклеотида, более предпочтительно
(a) по меньшей мере одну модификацию основания, предпочтительно выбранную из 2-тиоурацила, 2-тиотимина, 5-метилцитозина, 5-метилурацила, тимина, 2,6-диаминопурина, более предпочтительно, выбранную из 5-метилпиримидина и 2,6-диаминопурина; и/или
(b) по меньшей мере одну модификацию сахара, предпочтительно выбранную из 2'-O-метила, 2'-O-(2-метокси)этила, 2'-O-дезокси (ДНК), 2'-F, морфолино, мостикового нуклеотида или BNA, или олигонуклеотид содержит как мостиковые нуклеотиды, так и 2'-дезокси-модифицированные нуклеотиды (смешанные BNA/ДНК-меры), более предпочтительно модификация сахара представляет собой 2'-O-метил; и/или
(c) по меньшей мере одну модификацию основной цепи, предпочтительно выбранную из фосфоротиоата или фосфородиамидата, более предпочтительно модификация основной цепи представляет собой фосфоротиоат.
Таким образом, олигонуклеотид согласно этому варианту осуществления изобретения содержит модификацию основания (a) и отсутствие модификации сахара (b) и отсутствие модификации основной цепи (c). Другой предпочтительный олигонуклеотид согласно этому аспекту изобретения включает модификацию сахара (b) и отсутствие модификации основания (a) и отсутствие модификации основной цепи (c). Другой предпочтительный олигонуклеотид согласно этому аспекту изобретения содержит модификацию основной цепи (c) и отсутствие модификации основания (a) и отсутствие модификации сахара (b). Также предполагается, что настоящее изобретение охватывает олигонуклеотиды, не имеющие ни одной из упомянутых выше модификаций, а также олигонуклеотиды, содержащие две, т.е. (a) и (b), (a) и (c) и/или (b) и (c), или все три из модификаций (a), (b) и (c), как определено выше.
В предпочтительном варианте осуществления олигонуклеотид по изобретению модифицирован по всей его длине одной или несколькими одинаковыми модификациями, выбранными из (a) одной из модификаций оснований; и/или (b) одной из модификаций сахара; и/или (c) одной из модификаций основной цепи.
С появлением технологии имитации нуклеиновых кислот стало возможным получать молекулы, которые обладают сходными, предпочтительно одинаковыми по типу характеристиками гибридизации, но не обязательно сходными количественными характеристиками, с самой нуклеиновой кислотой. Такие функциональные эквиваленты, безусловно, также являются пригодными для применения в рамках изобретения.
В другом предпочтительном варианте осуществления олигонуклеотид содержит инозин, гипоксантин, универсальное основание, вырожденное основание и/или нуклеозид или нуклеотид, содержащие основание, способное образовывать качающуюся пару оснований или ее функциональный эквивалент. Применение инозина (гипоксантина) и/или универсального основания и/или вырожденного основания и/или нуклеотида, содержащего основание, способное образовывать качающуюся пару оснований, в олигонуклеотиде по изобретению, является высоко перспективным, как пояснено ниже. Инозин, например, является известным модифицированным основанием, которое может образовывать пару с тремя основаниями: урацил, аденин и цитозин. Инозин представляет собой нуклеозид, который образуется, когда гипоксантин связан с кольцом рибозы (также известным как рибофураноза) через β [бета]-N9-гликозидную связь. Инозин (I) обычно встречается в тРНК, и он необходим для надлежащей трансляции генетического кода в качающихся парах оснований. Качающаяся пара оснований может существовать между G и U, или между I с одной стороны и U, A или C с другой стороны. Это является фундаментальным при образовании вторичной структуры РНК. Ее термодинамическая стабильность сравнима с термодинамической стабильностью пары оснований Уотсона-Крика. Генетический код исправляет несоответствия в количестве аминокислот (20) для триплетных кодонов (64), с использованием модифицированных пар оснований в первом основании антикодона.
Первое преимущество использования инозина (гипоксантина) и/или универсального основания и/или вырожденного основания и/или нуклеотида, содержащего основание, способное образовывать качающуюся пару оснований, в олигонуклеотиде по изобретению, позволяет конструировать олигонуклеотид, который способен связываться с областью первого экзона из пре-мРНК и способен связываться с областью второго экзона в той же пре-мРНК, где указанная область из указанного второго экзона обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона. Иными словами, присутствие инозина (гипоксантина) и/или универсального основания и/или вырожденного основания и/или нуклеотида, содержащего основание, способное образовывать качающуюся пару оснований, в олигонуклеотиде по изобретению, позволяет связывание указанного олигонуклеотида с областью первого экзона и с областью второго экзона указанной пре-мРНК.
Второе преимущество использования инозина (гипоксантина) и/или универсального основания и/или вырожденного основания и/или нуклеотида, содержащего основание, способное образовывать качающуюся пару оснований, в олигонуклеотиде по изобретению, позволяет конструировать олигонуклеотид, который охватывает однонуклеотидный полиморфизм (SNP), без опасения, что полиморфизм нарушит эффективность отжига олигонуклеотида. Таким образом, в рамках изобретения использование такого основания позволяет конструирование олигонуклеотида, который можно использовать для индивидуума, имеющего SNP в участке пре-мРНК, на который нацелен олигонуклеотид по изобретению.
Третье преимущество использования инозина (гипоксантина) и/или универсального основания и/или вырожденного основания и/или нуклеотида, содержащего основание, способное образовывать качающуюся пару оснований, в олигонуклеотиде по изобретению, представляет собой случай, когда указанный олигонуклеотид в норме содержит CpG, если он сконструирован как являющийся комплементарным части первого экзона пре-мРНК, как определено в настоящем описании. Присутствие CpG в олигонуклеотиде обычно ассоциировано с увеличенной иммуногенностью указанного олигонуклеотида (Dorn A. and Kippenberger S.). Эта увеличенная иммуногенность является нежелательной, поскольку она может индуцировать разрушение мышечных волокон. Ожидается, что замена гуанина инозином в одном, двух или более CpG в указанном олигонуклеотиде обеспечит олигонуклеотид со сниженным и/или приемлемым уровнем иммуногенности. Иммуногенность можно оценивать в модели на животных путем оценки присутствия клеток CD4+ и/или CD8+ и/или инфильтрации воспалительных мононуклеоцитов в биоптате мышц указанного животного. Иммуногенность также можно оценивать в крови животного или человека, подвергаемого лечению олигонуклеотидом по изобретению, путем обнаружения присутствия нейтрализующего антитела и/или антитела, распознающего указанный олигонуклеотид, с использованием стандартного иммуноанализа, известного квалифицированному специалисту. Увеличение иммуногенности предпочтительно соответствует поддающемуся обнаружению увеличению по меньшей мере одного из этих типов клеток по сравнению с количеством каждого типа клеток в соответствующем биоптате мышц животного до обработки или лечения соответствующим олигонуклеотидом, имеющим по меньшей мере один инозин (гипоксантин) и/или универсальное основание и/или вырожденное основание и/или нуклеотид, содержащий основание, способное образовывать качающуюся пару оснований. Альтернативно, увеличение иммуногенности можно оценивать путем обнаружения присутствия или увеличения количества нейтрализующего антитела или антитела, распознающего указанный олигонуклеотид, с использованием стандартного иммуноанализа. Снижение иммуногенности предпочтительно соответствует поддающемуся обнаружению снижению по меньшей мере одного из этих типов клеток по сравнению с количеством соответствующего типа клеток в соответствующем биоптате мышц животного до лечения или после лечения соответствующим олигонуклеотидом, не имеющим инозина (гипоксантина) и/или универсального основания и/или вырожденного основания и/или нуклеотида, содержащего основание, способное образовывать качающуюся пару оснований. Альтернативно, снижение иммуногенности можно оценивать по отсутствию или снижению количества указанного соединения и/или нейтрализующих антител с использованием стандартного иммуноанализа.
Четвертым преимуществом использования инозина (гипоксантина) и/или универсального основания и/или вырожденного основания и/или нуклеотида, содержащего основание, способное образовывать качающуюся пару оснований, в олигонуклеотиде по изобретению, является избегание или снижение потенциальной мультимеризации или агрегации олигонуклеотидов. Например, известно, что олигонуклеотид, содержащий мотив G-квартет, имеет тенденцию к образованию квадруплекса, мультимера или агрегата, образованного хугстиновским спариванием оснований четырех одноцепочечных олигонуклеотидов (Cheng A.J. и Van Dyke M.W.), что, безусловно, нежелательно: ожидается, что в результате этого эффективность олигонуклеотида снизится. Мультимеризацию или агрегацию предпочтительно оценивают стандартными способами полиакриламидного неденатурирующего гель-электрофореза, известными квалифицированному специалисту. В предпочтительном варианте осуществления менее 20% или 15%, 10%, 7%, 5% или менее от общего количества олигонуклеотида по изобретению обладает способностью к мультимеризации или агрегации, оцениваемой с использованием анализа, упомянутого выше.
Пятым преимуществом использования инозина (гипоксантина) и/или универсального основания и/или вырожденного основания и/или нуклеотида, содержащего основание, способное образовывать качающуюся пару оснований, в олигонуклеотиде по изобретению, таким образом, также является избегание квадруплексных структур, которая ассоциирована с антитромботической активностью (Macaya R.F., et al.), а также со связыванием с и ингибированием фагоцитарного рецептора макрофагов (Suzuki K., et al.).
Шестым преимуществом использования инозина (гипоксантина) и/или универсального основания и/или вырожденного основания и/или нуклеотида, содержащего основание, способное образовывать качающуюся пару оснований, в олигонуклеотиде по изобретению, является возможность конструирования олигонуклеотида с улучшенной кинетикой связывания РНК и/или термодинамическими свойствами. Кинетика связывания РНК и/или термодинамические свойства по меньшей мере частично определяются температурой плавления олигонуклеотида (Tm; вычисляемая с помощью калькулятора свойств олигонуклеотидов (http://www.unc.edu/~cail/biotool/oligo/index.html) для одноцепочечной РНК с использованием базовой Tm и модели ближайшего соседа) и/или свободной энергией комплекса AON и экзона-мишени (с использованием RNA structure версии 4.5). Если Tm является чрезмерно высокой, ожидается, что олигонуклеотид будет менее специфичным. Приемлемая Tm и свободная энергия зависят от последовательности олигонуклеотида. Таким образом, трудно привести предпочтительные диапазоны для каждого из этих параметров. Приемлемая Tm может находиться в диапазоне от 35 до 85°C, и приемлемая свободная энергия может находиться в диапазоне от 15 до 45 ккал/моль.
В зависимости от длины, олигонуклеотид по настоящему изобретению может содержать по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 остатков инозина (гипоксантина) и/или универсальных оснований и/или вырожденных оснований и/или нуклеотидов, содержащих основание, способное образовывать качающуюся пару оснований, или их функциональные эквиваленты.
Предпочтительно, указанный олигонуклеотид по изобретению содержит РНК, поскольку дуплексы РНК/РНК являются высокостабильными. Является предпочтительным, чтобы РНК-олигонуклеотид содержал модификацию, обеспечивающую РНК с дополнительным свойством, например, устойчивостью к эндонуклеазам, экзонуклеазам и РНК-азе H, дополнительной прочностью гибридизации, увеличенной стабильностью (например, в жидкостях организма), увеличенной или сниженной пластичностью, сниженной токсичностью, увеличенным внутриклеточным транспортом, тканевой специфичностью и т.д. Предпочтительные модификации описаны выше.
Таким образом, один вариант осуществления относится к олигонуклеотиду, который содержит по меньшей мере одну модификацию. Предпочтительный модифицированный олигонуклеотид является полностью 2'-O-метил-модифицированным. В одном варианте осуществления изобретения олигонуклеотид содержит или состоит из гибридного олигонуклеотида, содержащего модификацию 2'-O-метил-фосфоротиоатным олигорибонуклеотидом и модификацию мостиковой нуклеиновой кислотой (BNA, как проиллюстрировано выше). В другом варианте осуществления изобретения олигонуклеотид содержит или состоит из гибридного олигонуклеотида, содержащего модификацию 2'-O-метоксиэтилфосфоротиоатом и мостиковую нуклеиновую кислоту (BNA, как проиллюстрировано выше). В другом варианте осуществления изобретения олигонуклеотид содержит или состоит из гибридного олигонуклеотида, содержащего модификацию мостиковой нуклеиновой кислотой (BNA, как проиллюстрировано выше) и модификацию олигодезоксирибонуклеотидом. Эта конкретная комбинация обладает лучшей специфичностью последовательности по сравнению с эквивалентом, состоящим только из мостиковой нуклеиновой кислоты, и обладает увеличенной эффективностью по сравнению с олигонуклеотидом, состоящим из модификации 2'-O-метилфосфоротиоатным олиго(дезокси)рибонуклеотидом.
Соединение, как описано в рамках изобретения, предпочтительно может обладать ионогенными группами. Ионогенные группы могут представлять собой основания или кислоты, и они могут быть заряженными или нейтральными. Ионогенные группы могут присутствовать в качестве пары ионов с соответствующим противоионом, который имеет противоположный заряд(ы). Примерами катионных противоионов являются натрий, калий, цезий, Tris, литий, кальций, магний, триалкиламмоний, триэтиламмоний и тетраалкиламмоний. Примерами анионных противоионов являются хлорид, бромид, йодид, лактат, мезилат, ацетат, трифторацетат, дихлорацетат и цитрат. Примеры противоионов описаны [например, Kumar L, которая включена в настоящее описание в качестве ссылки в полном объеме]. Примеры применения двух- или трехвалентных противоионов, особенно Ca2+, описаны в качестве дополнительных положительных характеристик к определенным олигонуклеотидам в патентной заявке США 2012046348 (Replicor), которая включена в качестве ссылки в полном объеме. Предпочтительные двухвалентные или трехвалентные противоионы выбирают из следующего перечня или группы: кальций, магний, кобальт, железо, марганец, барий, никель, медь и цинк. Предпочтительным двухвалентным противоионом является кальций. Таким образом, настоящее изобретение охватывает приготовление, получение и применение композиции, содержащей олигонуклеотид по изобретению и любой другой противоион, определенный выше, предпочтительно кальций.
Такой способ получения указанной композиции, содержащей указанный олигонуклеотид и указанный противоион, предпочтительно кальций, может быть следующим: олигонуклеотид по изобретению можно растворять в фармацевтически приемлемом водном эксципиенте, и постепенно к растворенному олигонуклеотиду можно добавлять раствор, содержащий указанный противоион, чтобы хелатный комплекс олигонуклеотида оставался растворимым.
Таким образом, в предпочтительном варианте осуществления олигонуклеотид по изобретению приводят в контакт с композицией, содержащей такой противоион, предпочтительно Ca2+, для формирования хелатного комплекса олигонуклеотида, содержащего два или более идентичных олигонуклеотидов, связанных таким противоионом, как определено в настоящем описании. Композиция, содержащая хелатный комплекс олигонуклеотида, содержащий два или более идентичных олигонуклеотида, связанных противоионом, предпочтительно кальцием, таким образом, охватывается настоящим изобретением.
Способ конструирования олигонуклеотида
Таким образом, в следующем аспекте изобретения предусматривается способ конструирования олигонуклеотида, где указанный способ приводит к олигонуклеотиду, как определено выше.
Этот способ включает следующие стадии:
(a) идентификация комбинации в рамке считывания первого и второго экзона в одной и той же пре-мРНК, где область указанного второго экзона обладает по меньшей мере 50% идентичностью с областью указанного первого экзона;
(b) конструирование олигонуклеотида, который способен связываться с указанной областью указанного первого экзона и указанной областью указанного второго экзона; и
(c) где указанное связывание приводит к пропусканию указанного первого экзона и указанного второго экзона, предпочтительно к пропусканию мультиэкзонного участка, начинающегося указанным первым экзоном и охватывающего один или несколько экзонов, присутствующих между указанным первым и указанным вторым экзонами, и, в лучшем случае, к пропусканию всего участка экзонов между указанным первым и указанным вторым экзонами.
На стадии b) такого способа указанный олигонуклеотид предпочтительно конструируют так, чтобы его связывание препятствовало по меньшей мере одной регулирующей сплайсинг последовательности в указанных областях указанного первого и/или второго экзонов в указанной пре-мРНК.
Альтернативно или в комбинации с препятствованием регулирующей сплайсинг последовательности, связывание указанного олигонуклеотида предпочтительно препятствует вторичной структуре, охватывающей по меньшей мере указанный первый и/или указанный второй экзоны в указанной пре-мРНК.
Олигонуклеотид, получаемый этим способом, способен индуцировать пропускание указанных первого и второго экзонов в указанной пре-мРНК. Предпочтительно индуцируется пропускание дополнительного экзона(ов), где указанный дополнительный экзон(ы) предпочтительно располагается между указанным первым и указанным вторым экзонами, и где полученный мРНК-транскрипт находится в рамке считывания. Олигонуклеотид предпочтительно способен индуцировать пропускание всего участка экзонов между указанным первым экзоном и указанным вторым экзоном. В одном варианте осуществления указанные области указанного первого экзона и указанного второго экзона содержат регулирующий сплайсинг элемент, так что связывание указанного олигонуклеотида способно препятствовать по меньшей мере одной регулирующей сплайсинг последовательности в указанных областях указанных первого и второго экзонов. Также предусматривается, что связывание указанного олигонуклеотида препятствует вторичной структуре, охватывающей по меньшей мере указанный первый и/или указанный второй экзоны в указанной пре-мРНК. Предпочтительная регулирующая сплайсинг последовательность содержит участок связывания серин-аргининового (SR) белка, энхансер сплайсинга экзонов (ESE), последовательность распознавания экзонов (ERS) и/или сайленсер сплайсинга экзонов (ESS).
Каждый признак этого способа уже определен в настоящем описании или известен квалифицированному специалисту.
Предпочтительные олигонуклеотиды
Предпочтительно, олигонуклеотид по изобретению предназначен для применения в качестве лекарственного средства, более предпочтительно, указанное соединение предназначено для применения в модулирующих РНК терапевтических средствах. Более предпочтительно, указанное модулирование РНК приводит к коррекции нарушенной транскрипционной рамки считывания и/или к восстановлению экспрессии желаемого или ожидаемого белка. Таким образом, способ по изобретению и олигонуклеотид по изобретению можно в принципе применять к любому заболеванию, связанному с присутствием нарушающей рамку считывания мутации, что приводит к аберрантному транскрипту и/или к отсутствию кодируемого белка и/или к присутствию аберрантного белка. В предпочтительном варианте осуществления способ по изобретению и олигонуклеотид по изобретению применяют к ассоциированным с заболеванием генам, содержащим повторяющиеся последовательности и, таким образом, области с относительно высокой идентичностью последовательности (т.е. по меньшей мере 50%, 60%, 70%, 80% идентичностью последовательности между областью второго экзона и областью первого экзона, как определено в настоящем описании). Неограничивающими примерами являются гены со спектрин-подобными повторами (поскольку ген DMD вовлечен в мышечную дистрофию Дюшенна, как описано в настоящем описании), Kelch-подобными повторами (как например, ген KLHL3, вовлеченный в семейную гиперкалиемическую гипертензию (Louis-Dit-Picard H et al.), ген KLHL6, вовлеченный в хронический лимфоцитарный лейкоз (Puente XS et al.), ген KLHL7, вовлеченный в пигментный ретинит (Friedman JS et al.), гены KLHL7/12, вовлеченные в синдром Шегрена (Uchida K et al.), ген KLHL9, вовлеченный в дистальную миопатию (Cirak S et al.), ген KLHL16 или GAN, вовлеченный в гигантскую аксональную невропатию (Bomont P et al.) , гены KLHL19 или KEAP1, вовлеченные в несколько злокачественных опухолей (Dhanoa BS et al.), ген KLHL20, вовлеченный в промиелоцитарный лейкоз (Dhanoa BS et al.) , или гены KLHL37 или ENC1, вовлеченные в опухоли головного мозга (Dhanoa BS et al.), FGF-подобными повторами, EGF-подобными повторами (как, например, ген NOTCH3, вовлеченный в CADASIL (Chabriat H et al.), гены SCUBE, вовлеченные в злокачественную опухоль или метаболическое заболевание костей, ген нейрексина-1 (NRXN1), вовлеченный в нейропсихиатрические нарушения и идиопатическую генерализованную эпилепсию (Moller RS et al.), ген Del-1, ген тенасцин-C (TNC), вовлеченный в атеросклероз и болезнь коронарных артерий (Mollie A et al.), ген THBS3 или ген фибриллина (FBN1), вовлеченный в синдром Марфана (Rantamaki T et al.), анкирин-подобными повторами (как, например, ген ANKRD1, вовлеченный в кардиомиопатию с дилятацией (Dubosq-Bidot L et al.), ген ANKRD2, ген ANKRD11, вовлеченные в синдром KBG (Sirmaci A et al.), ген ANKRD26, вовлеченный в тромбоцитопению (Noris P et al.) или диабет (Raciti GA et al.), ген ANKRD55, вовлеченный в рассеянный склероз (Alloza I et al.), или ген TRPV4, вовлеченный в дистальную спинальную мышечную атрофию (Fiorillo C et al.), HEAT-подобными повторами (как, например, ген, вовлеченный в болезнь Хантингтона), аннексин-подобными повторами, лейцин-богатыми повторами (как, например, гены NLRP2 и NLRP7, вовлеченные в идиопатическое привычное невынашивание беременности (Huang JY et al.)), ген LRRK2, вовлеченный в болезнь Паркинсона (Abeliovich A et al.), ген NLRP3, вовлеченный в болезнь Альцгеймера или менингит (Heneka MT et al.), ген NALP3, вовлеченный в почечную недостаточность (Knauf F et al.), или ген LRIG2, вовлеченный в урофациальный синдром (Stuart HM et al.), или доменами ингибитора сериновой протеазы (как, например, ген SPINK5, вовлеченный в синдром Незертона (Hovnanian A et al.), как описано в Andrade M.A. et al.
В контексте изобретения предпочтительной пре-мРНК или транскриптом является пре-мРНК или транскрипт дистрофина. Эта предпочтительная пре-мРНК или транскрипт предпочтительно являются человеческими. Заболевание, связанное с присутствием мутации, присутствующей в пре-мРНК дистрофина, представляет собой DMD или BMD, в зависимости от мутации. Предпочтительные комбинации и области первого и второго экзонов из пре-мРНК дистрофина, подлежащие применению в контексте изобретения, представлены в таблице 2.
В предпочтительном варианте осуществления соединение по изобретению используют для индукции пропускания экзона в клетке, в органе, в ткани и/или у пациента, предпочтительно у пациента с BMD или DMD или в клетке, органе, ткани, происходящих из указанного пациента. Пропускание экзона приводит к зрелой мРНК, которая не содержит пропущенного экзона, и, таким образом, когда указанный экзон кодирует аминокислоты, оно может приводить к экспрессии измененного, внутренне укороченного, но от частично до в большой степени функционального продукта дистрофина. Технология пропускания экзонов в настоящее время направлена на применение AON или транскрибирующих AON генных конструкций. Способы пропускания экзонов в настоящее время используют для борьбы с генетическими мышечными дистрофиями. Недавно авторами настоящего изобретения и другими исследователями были сообщены перспективные результаты в отношении терапии на основе индуцируемого AON пропускания экзонов, нацеленной на восстановление рамки считывания пре-мРНК дистрофина из мыши mdx и от пациентов с DMD (Heemskerk H., et al., Cirak S., et al., Goemans et al.). Путем направленного пропускания конкретного экзона тяжелый фенотип DMD (отсутствие функционального дистрофина) преобразуется в более мягкий фенотип BMD (экспрессия функционального или полуфункционального дистрофина). Пропускание экзона предпочтительно индуцируется связыванием AON, нацеленных на внутреннюю последовательность экзона.
Ниже изобретение иллюстрируется при помощи мутантной пре-мРНК дистрофина, где присутствуют первый и второй экзоны. Как определено в настоящем описании, пре-мРНК дистрофина предпочтительно означает пре-мРНК гена DMD, кодирующего белок дистрофина. Мутантная пре-мРНК дистрофина соответствует пре-мРНК пациента с BMD или DMD с мутацией по сравнению с пре-мРНК DMD дикого типа у здорового индивидуума, что приводит к (сниженным уровням) аберрантному белку (BMD), или к отсутствию функционального дистрофина (DMD). Пре-мРНК дистрофина также называют пре-мРНК DMD. Ген дистрофина также может быть называться геном DMD. Дистрофин и DMD могут использоваться в настоящем описании взаимозаменяемо.
Пациент предпочтительно означает пациента, имеющего DMD или BMD, как определено в настоящем описании ниже, или пациента, предрасположенного к развитию DMD или BMD вследствие его (или ее) генетического фона. В случае пациента с DMD, используемое соединение предпочтительно корректирует мутацию, присутствующую в гене DMD указанного пациента и, таким образом, предпочтительно образует белок дистрофина, сходный с белком дистрофина пациента с BMD: указанный белок предпочтительно представляет собой функциональный или полуфункциональный дистрофин, как определено в настоящем описании ниже. В случае пациента с BMD, антисмысловой олигонуклеотид по изобретению предпочтительно модифицирует мутацию, присутствующую в гене BMD указанного пациента и предпочтительно образует дистрофин, который является более функциональным, чем дистрофин, который исходно присутствует у указанного пациента с BMD.
Как определено в настоящем описании, функциональный дистрофин предпочтительно представляет собой дистрофин дикого типа, соответствующий белку, имеющему аминокислотную последовательность, как указано в SEQ ID NO: 1. Функциональный дистрофин предпочтительно представляет собой дистрофин, который имеет действующий связывающий домен в его N-концевой части (первые 240 аминокислот на N-конце), цистеин-богатый домен (аминокислоты с 3361 по 3685) и C-концевой домен (последние 325 аминокислот на C-конца), причем каждый из этих доменов присутствует в дистрофине дикого типа, как известно квалифицированному специалисту. Аминокислоты, указанные в настоящем описании, соответствуют аминокислотам дистрофина дикого типа, указанным в SEQ ID NO: 1. Иными словами, функциональный или полуфункциональный дистрофин представляет собой дистрофин, который проявляет по меньшей мере в некоторой степени активность дистрофина дикого типа. "По меньшей мере в некоторой степени" предпочтительно означает по меньшей мере 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95% или 100% соответствующей активности функционального дистрофина дикого типа. В этом контексте активность функционального дистрофина предпочтительно представляет собой связывание с актином и взаимодействие с ассоциированным с дистрофином гликопротеиновым комплексом (DGC) или (DAGC) (Ehmsen J et al.). Связывание дистрофина с актином и с комплексом DGC можно визуализировать либо посредством ко-иммунопреципитации с использованием экстрактов тотального белка, либо посредством иммунофлуоресцентного анализа поперечных срезов из биоптата мышцы, предположительно являющейся дистрофичной, как известно квалифицированному специалисту.
Индивидуумы, страдающие DMD, как правило, имеют мутацию в гене, кодирующем дистрофин, которая препятствует синтезу полного белка, например, преждевременный стоп-кодон препятствует синтезу C-конца. При BMD ген дистрофина также содержит мутацию, однако эта мутация не нарушает открытую рамку считывания и синтезируется C-конец. В результате образуется (полу)функциональный или функциональный белок дистрофин, который обладает сходным типом активности с белком дикого типа, хотя и не обязательно сходным уровнем активности. Геном индивидуума с BMD, как правило, кодирует белок дистрофин, содержащий N-концевую часть (первые 240 аминокислот на N-конце), цистеин-богатый домен (аминокислоты с 3361 по 3685) и C-концевой домен (последние 325 аминокислот на C-конце), однако в большинстве случаев его центральный домен в форме стержня может быть короче, чем у дистрофина дикого типа (Monaco A.P., et al.).
Пропускание экзонов для лечения DMD, как правило, относится к обходу преждевременного стоп-кодона в пре-мРНК путем пропускания экзона, фланкирующего или содержащего мутацию. Это позволяет коррекцию открытой рамки считывания и синтез внутренне укороченного белка дистрофина, но включающего C-конец. В предпочтительном варианте осуществления у индивидуума, имеющего DMD и подвергаемого лечению олигонуклеотидом по изобретению, синтезируется дистрофин, который проявляет, по меньшей мере в некоторой степени, сходную активность с дистрофином дикого типа. Более предпочтительно, если указанный индивидуум является пациентом с DMD или предположительно является пациентом с DMD, (полу)функциональный дистрофин представляет собой дистрофин индивидуума, имеющего BMD: как правило, указанный дистрофин способен взаимодействовать как с актином, так и с DGC или DAGC (Ehmsen J., et al., Monaco A.P., et al.).
Центральный стержневой домен дистрофина дикого типа содержит 24 спектрин-подобных повтора (Ehmsen J., et al.). Во многих случаях центральный стержневой домен в BMD-подобных белках является более коротким, чем в дистрофине дикого типа (Monaco A.P., et al.). Например, центральный стержневой домен дистрофина, как предусматривается в настоящем описании, может содержать от 5 до 23, от 10 до 22 или от 12 до 18 спектрин-подобных повторов при условии, что он может связываться с актином и с DGC.
Смягчение одного или нескольких симптомов DMD или BMD у индивидуума с использованием соединения по изобретению можно оценивать с помощью любого из следующих анализов: продление времени до прекращения хождения, увеличение мышечной силы, повышение способности поднимать тяжести, улучшение времени вставания с пола, улучшение времени ходьбы на девять метров, улучшение времени подъема на четыре ступеньки, улучшение степени функции ног, улучшение легочной функции, улучшение сердечной функции, улучшение качества жизни. Каждый из этих анализов известен квалифицированному специалисту. В качестве примера, в публикации Manzur et al. (Manzur A.Y. et al.) приводится обширное пояснение каждого из этих анализов. Для каждого из этих анализов, когда выявляют поддающееся обнаружению улучшение или продление параметра, измеряемого в анализе, это предпочтительно означает, что один или несколько симптомов DMD или BMD смягчились у индивидуума с использованием соединения по изобретению. Поддающееся обнаружению улучшение или продление предпочтительно представляет собой статистически значимое улучшение или продление, как описано в Hodgetts et al. (Hodgetts S., et al.). Альтернативно, смягчение одного или нескольких симптомов DMD или BMD можно оценивать посредством измерения улучшения функции, целостности и/или выживаемости мышечных волокон. В предпочтительном способе один или несколько симптомов пациента с DMD или BMD смягчается и/или одна или несколько характеристик одной или нескольких мышечных клеток от пациента с DMD или BMD улучшается. Такие симптомы или характеристики можно оценивать на клеточном, тканевом уровне или у самого пациента.
Смягчение одной или нескольких характеристик мышечной клетки от пациента с DMD или BMD можно оценивать с помощью любого из следующих анализов на миогенной клетке или мышечной клетке от этого пациента: сниженный захват кальция мышечными клетками, сниженный синтез коллагена, измененная морфология, измененный биосинтез липидов, сниженный окислительный стресс и/или улучшенная функция, целостность и/или выживаемость мышечных волокон. Эти параметры обычно оценивают с использованием иммунофлуоресцентных и/или гистохимических анализов поперечных срезов биоптатов мышц.
Улучшение функции, целостности и/или выживаемости мышечных волокон можно оценивать с использованием по меньшей мере одного из следующих анализов: поддающееся обнаружению снижение уровня креатинкиназы в крови, поддающееся обнаружению снижение некроза мышечных волокон в поперечном срезе биоптата мышцы, предположительно являющейся дистрофической, и/или поддающееся обнаружению повышение гомогенности диаметра мышечных волокон в поперечном срезе биоптата мышцы, предположительно являющейся дистрофической. Каждый из этих анализов известен квалифицированному специалисту.
Креатинкиназу можно выявлять в крови, как описано в Hodgetts et al. (Hodgetts S., et al.). Поддающееся обнаружению снижение креатинкиназы может означать снижение на 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или более по сравнению с концентрацией креатинкиназы у того же пациента с DMD или BMD до лечения.
Поддающееся обнаружению снижение некроза мышечных волокон предпочтительно оценивают в биоптате мышц, более предпочтительно, как описано в Hodgetts et al. (Hodgetts S., et al.), с использованием поперечных срезов биоптатов. Поддающееся обнаружению снижение некроза может представлять собой снижение, составляющее 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или более от площади, где идентифицирован некроз с использованием поперечных срезов биоптатов. Снижение измеряют путем сравнения с некрозом, как оценивают у того же пациента с DMD или BMD до лечения.
Поддающееся обнаружению повышение гомогенности диаметра мышечных волокон предпочтительно оценивают в поперечном срезе биоптата, более предпочтительно, как описано в Hodgetts et al. (Hodgetts S., et al.). Увеличение измеряют путем сравнения с гомогенностью диаметра мышечного волокна у того же пациента с DMD или BMD до лечения.
Предпочтительно, олигонуклеотид по изобретению обеспечивает у указанного индивидуума (более высокие уровни) функциональный и/или (полу)функциональный белок дистрофин (как для DMD, так и для BMD) и/или способен, по меньшей мере частично, снижать продукцию аберрантного белка дистрофина у указанного индивидуума. В этом контексте, функциональный и/или полуфункциональный дистрофин может означать, что может образовываться несколько форм функционального и/или полуфункционального дистрофина. Этого можно ожидать, когда область с по меньшей мере 50% идентичностью между первым и вторым экзонами перекрывается с одной или несколькими другими областями с по меньшей мере 50% идентичностью между другими первым и вторым экзонами, и где указанный олигонуклеотид способен связываться с указанной перекрывающейся частью, так что несколько различных участков экзонов пропускается и продуцируется несколько различных транскриптов в рамке считывания. Эта ситуация иллюстрируется в примере 4, где один единственный олигонуклеотид по изобретению (PS816; SEQ ID NO: 1679) способен индуцировать продукцию нескольких транскриптов в рамке считывания, все из которых имеют общий экзон 10 в качестве первого экзона, и где второй экзон может представлять собой экзон 13, 14, 15, 18, 20, 27, 30, 31, 32, 35, 42, 44, 47, 48 или 55.
Более высокие уровни относятся к увеличению уровня функционального и/или (полу)функционального белка дистрофина по сравнению с соответствующим уровнем функционального и/или (полу)функционального белка дистрофина у пациента до начала лечения олигонуклеотидом по изобретению. Уровень указанного функционального и/или (полу)функционального белка дистрофина предпочтительно оценивают с использованием иммунофлуоресцентного анализа или анализа с использованием вестерн-блоттинга (белок).
Снижение продукции аберрантной мРНК дистрофина или аберрантного белка дистрофина предпочтительно означает, что 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10%, 5% или менее от первоначального количества аберрантной мРНК дистрофина или аберрантного белка дистрофина все еще поддается обнаружению с помощью ОТ-ПЦР (мРНК) или иммунофлуоресцентного анализа или анализа с использованием вестерн-блоттинга (белок). Аберрантную мРНК или белок дистрофина также обозначают в настоящем описании как менее функциональную (по сравнению с функциональным белком дистрофина дикого типа, как определено в настоящем описании ранее) или нефункциональную мРНК или белок дистрофина. Нефункциональный белок дистрофина предпочтительно представляет собой белок дистрофина, который не способен связывать актин и/или представителей белкового комплекса DGC. Нефункциональный белок дистрофина или мРНК дистрофина, как правило, не обладает или не кодирует белок дистрофина с интактным C-концом белка. В предпочтительном варианте осуществления используют способ пропускания экзонов (также называемый модулированием РНК или переключением сплайсинга).
Увеличение продукции функциональной и/или полуфункциональной мРНК и/или белка дистрофина предпочтительно означает, что такая функциональная и/или полуфункциональная мРНК и/или белок дистрофина поддаются обнаружению или увеличены по меньшей мере на 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или более по сравнению с выявляемым количеством указанной мРНК и/или белка в начале лечения. Указанное выявление можно осуществлять с использованием ОТ-ПЦР (мРНК) или иммунофлуоресцентного анализа или анализа с использованием вестерн-блоттинга (белок).
В другом варианте осуществления соединение по изобретению обеспечивает у указанного индивидуума функциональный или полуфункциональный белок дистрофина. Этот функциональный или полуфункциональный белок или мРНК дистрофина можно выявлять в качестве аберрантного белка или мРНК дистрофина, как пояснено в настоящем описании выше.
Посредством направленного совместного пропускания указанных двух экзонов (указанный первый и указанный второй экзоны) можно индуцировать пропускание дополнительного экзона(ов), где указанный дополнительный экзон(ы) предпочтительно расположен между указанным первым и указанным вторым экзонами, и где полученный транскрипт дистрофина находится в рамке считывания (предпочтительно, как в таблице 1 или 6), фенотип DMD или тяжелой BMD преобразуется в более мягкий фенотип BMD или даже бессимптомный фенотип. Совместное пропускание указанных двух экзонов предпочтительно индуцируется связыванием олигонуклеотида с областью первого экзона в пре-мРНК дистрофина и связыванием олигонуклеотида с областью второго экзона в пре-мРНК дистрофина, где указанная область указанного второго экзона обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона. Предпочтительно, указанный первый и указанный второй экзоны дистрофина и области идентичности в них представлены в таблице 2 или 6. Указанный олигонуклеотид предпочтительно не обладает перекрыванием с последовательностями не экзонов. Указанный олигонуклеотид предпочтительно не перекрывается с участками сплайсинга, по меньшей мере не в той степени, в какой они присутствуют в интроне. Указанный олигонуклеотид, направленный на внутреннюю последовательность экзона, предпочтительно не содержит последовательность, обратно комплементарную соседнему интрону. Способ пропускания экзонов предпочтительно используют так, что отсутствие указанных двух экзонов, предпочтительно дополнительного экзона(ов), более предпочтительно расположенного между указанным первым и указанным вторым экзонами, из мРНК, продуцированной с пре-мРНК DMD, образует кодирующую область для (более высокой) экспрессии более (полу)функционального - хотя и более короткого - белка дистрофина. В этом контексте (как правило, у пациента с BMD), ингибирование включения указанных двух экзонов, предпочтительно дополнительного экзона(ов), расположенных между указанным первым и указанным вторым экзонами, предпочтительно означает, что:
- уровень исходной аберрантной (менее функциональной) мРНК дистрофина снижен по меньшей мере на 5% при оценке способом ОТ-ПЦР, или уровень соответствующего аберрантного белка дистрофина снижается по меньшей мере на 2% при оценке с помощью иммунофлуоресцентного анализа или анализа с использованием вестерн-блоттинга с использованием антител против дистрофина; и/или
- транскрипт в рамке считывания, кодирующий полуфункциональный или функциональный белок дистрофин, поддается обнаружению или его уровень возрастает по меньшей мере на 5% при оценке способом ОТ-ПЦР (уровень мРНК) или по меньшей мере на 2% при оценке с помощью иммунофлуоресцентного анализа или вестерн-блоттинга с использованием антител против дистрофина (белковый уровень).
Снижение аберрантного менее функционального или нефункционального белка дистрофина предпочтительно составляет по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или 100%, и предпочтительно оно происходит одновременно или параллельно выявлению или увеличенной продукции более функционального или полуфункционального транскрипта или белка дистрофина. В этом контексте (как правило, у пациента с DMD), ингибирование включения указанных двух экзонов, предпочтительно дополнительного экзона(ов), расположенного между указанным первым и указанным вторым экзонами, предпочтительно означает, что у указанного индивидуума обеспечивается (более высокий уровень) более функциональный или (полу)функциональный белок или мРНК дистрофина.
После того, как у пациента с DMD обеспечивают (более высокие уровни) (более) функциональный или полуфункциональный белок дистрофина, причина DMD, по меньшей мере частично, смягчается. Таким образом, можно ожидать, что симптомы DMD в достаточной степени снижаются. Кроме того, настоящее изобретение относится к пониманию того, что пропускание всего участка из по меньшей мере двух экзонов дистрофина в пре-мРНК, содержащей указанные экзоны, индуцируется или усиливается при использовании единственного олигонуклеотида, направленного на (или способного связываться с, или гибридизоваться с, или являющегося обратно комплементарным или способного нацеливаться на) оба из внешних экзона указанного участка. В настоящей заявке, в предпочтительном варианте осуществления олигонуклеотид по изобретению, таким образом, по меньшей мере на 80% обратно комплементарен указанной области указанного первого экзона и по меньшей мере на 45% обратно комплементарен указанной области указанного второго экзона, как определено в настоящем описании, где указанные первый и второй экзоны соответствуют наружным экзонам участка экзонов, подлежащего пропусканию. Более предпочтительно, указанный олигонуклеотид по меньшей мере на 85%, 90% 95% или 100% обратно комплементарен указанной области указанного первого экзона и по меньшей мере на 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% или 100% обратно комплементарен указанной области указанного второго экзона. Увеличенная частота пропускания также увеличивает уровень более (полу)функционального белка дистрофина, продуцированного в мышечной клетки индивидуума с DMD или BMD.
Олигонуклеотид в соответствии с настоящим изобретением, который предпочтительно используют, предпочтительно является обратно комплементарным или способен связываться, или гибридизоваться, или нацеливаться на область первого экзона дистрофина, причем указанная область имеет по меньшей мере 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200 или более нуклеотидов, предпочтительно является обратно комплементарной или способна связываться, или гибридизоваться, или нацеливаться на область второго экзона дистрофина, причем указанная область имеет по меньшей мере 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200 или более нуклеотидов, где указанная область указанного второго экзона в той же пре-мРНК обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона.
В контексте изобретения олигонуклеотид может содержать или состоять из функционального эквивалента олигонуклеотида. Функциональный эквивалент олигонуклеотида предпочтительно означает олигонуклеотид, как определено в настоящем описании, где один или несколько нуклеотидов заменены, и где активность указанного функционального эквивалента сохраняется по меньшей мере в некоторой степени. Предпочтительно, активность указанного соединения, содержащего функциональный эквивалент олигонуклеотида, представляет собой обеспечение функционального или полуфункционального белка дистрофина. Указанную активность указанного соединения, содержащего функциональный эквивалент олигонуклеотида, таким образом, предпочтительно оценивают путем количественного определения функционального или полуфункционального белка дистрофина. Функциональный или полуфункциональный дистрофин предпочтительно определяют в рамках настоящего изобретения как представляющий собой дистрофин, способный связываться с актином и представителями комплекса белков DGC и поддерживать структуру и гибкость мембран мышечных волокон. Оценка указанной активности соединения, содержащего функциональный олигонуклеотид или эквивалент, предпочтительно включает ОТ-ПЦР (для обнаружения пропускания экзона на уровне РНК) и/или иммунофлуоресценцию или анализ с использованием вестерн-блоттинга (для обнаружения экспрессии и локализации белка). Указанная активность предпочтительно сохраняется, по меньшей мере в некоторой степени, когда она соответствует по меньшей мере 50%, или по меньшей мере 60%, или по меньшей мере 70%, или по меньшей мере 80%, или по меньшей мере 90%, или по меньшей мере 95% или более соответствующей активности указанного соединения, содержащего олигонуклеотид, из которого происходит функциональный эквивалент. На протяжении настоящей заявки, когда используют слово "олигонуклеотид", оно может быть заменено его функциональным эквивалентом, как определено в настоящем описании.
Таким образом, применение олигонуклеотида или его функционального эквивалента, содержащего или состоящего из последовательности:
которая способна связываться с, нацеливаться на, гибридизоваться с и/или является обратно комплементарной области первого экзона дистрофина,
которая способна связываться с, нацеливаться на, гибридизоваться с и/или является обратно комплементарной области второго экзона дистрофина,
и
где указанная область второго экзона в пре-мРНК дистрофина обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона, проявляет терапевтические результаты в отношении DMD посредством:
- смягчения одного или нескольких симптомов DMD или BMD; и/или
- смягчения одной или нескольких характеристик мышечной клетки от пациента; и/или
- обеспечения у указанного индивидуума функционального или полуфункционального белка дистрофина; и/или
- по меньшей мере частичного снижения продукции аберрантного белка дистрофина у указанного индивидуума.
Каждый из этих признаков уже определен в настоящем описании.
Предпочтительно, олигонуклеотид содержит или состоит из последовательности, которая способна связываться с, нацеливаться на, гибридизоваться с и/или является обратно комплементарной области первого экзона DMD или дистрофина, где область второго экзона DMD или дистрофина в той же пре-мРНК обладает по меньшей мере 50% идентичностью с указанной областью указанного первого экзона. Указанные первый и второй экзоны предпочтительно выбирают из группы экзонов, включающей экзоны с 8 по 60, где участок экзонов, начинающийся с первого экзона и содержащий второй экзон в качестве последнего экзона, участок экзонов, начинающийся первым экзоном и содержащий второй экзон в качестве последнего экзона, когда он пропускается, обеспечивает транскрипт в рамке считывания. Предпочтительные комбинации экзонов в рамке считывания в мРНК дистрофина приведены в таблице 1, 2 или 6.
Без связи с какой-либо теорией, идентичность первого и второго экзонов дистрофина можно определять с помощью одного или нескольких из следующих аспектов. В одном варианте осуществления один или несколько интронов, присутствующих между первым экзоном и вторым экзоном, не являются чрезмерно большими. В этом контексте, чрезмерно большой интрон в гене дистрофина может составлять 70, 80, 90, 100, 200 т.п.н. или более; например, интрон 1 (~83 т.п.н.), интрон 2 (~170 т.п.н.), интрон 7 (~110 т.п.н.), интрон 43 (~70 т.п.н.), интрон 44 (~248 т.п.н.), интрон 55 (~119 т.п.н.) или интрон 60 (~96 т.п.н.). Кроме того, в следующем варианте осуществления может предусматриваться пример уже пациента с BMD, у которого экспрессируется укороченный белок дистрофин, где этот первый, этот второй экзон и участок экзонов от указанного первого экзона до указанного второго экзона удалены. Другим критерием может быть относительно большая применимость пропущенных экзонов для объединенных субпопуляций пациентов с DMD (и/или BMD) с конкретными соответствующими мутациями.
В предпочтительном варианте осуществления пропускается участок экзонов DMD в рамке считывания (более предпочтительно, полностью пропускается в одном транскрипте), где наружные экзоны определяются первым и вторым экзонами следующим образом:
- первый экзон представляет собой экзон 8, и второй экзон представляет собой экзон 19 (применимо для ~7% пациентов с DMD),
- первый экзон представляет собой экзон 9, и второй экзон представляет собой экзон 22 (применимо для ~11% пациентов с DMD),
- первый экзон представляет собой экзон 9, и второй экзон представляет собой экзон 30 (применимо для ~14% пациентов с DMD),
- первый экзон представляет собой экзон 10, и второй экзон представляет собой экзон 18 (применимо для ~5% пациентов с DMD),
- первый экзон представляет собой экзон 10 и второй экзон представляет собой экзон 30 (применимо для ~13% пациентов с DMD),
- первый экзон представляет собой экзон 10, и второй экзон представляет собой экзон 42 (применимо для ~16% пациентов с DMD),
- первый экзон представляет собой экзон 10, и второй экзон представляет собой экзон 47 (применимо для ~29% пациентов с DMD),
- первый экзон представляет собой экзон 10, и второй экзон представляет собой экзон 57 (применимо для ~72% пациентов с DMD),
- первый экзон представляет собой экзон 10, и второй экзон представляет собой экзон 60 (применимо для ~72% пациентов с DMD),
- первый экзон представляет собой экзон 11, и второй экзон представляет собой экзон 23 (применимо для ~8% пациентов с DMD),
- первый экзон представляет собой экзон 13 и второй экзон представляет собой экзон 30 (применимо для ~10% пациентов с DMD),
- первый экзон представляет собой экзон 23, и второй экзон представляет собой экзон 42 (применимо для ~7% пациентов с DMD),
- первый экзон представляет собой экзон 34 и второй экзон представляет собой экзон 53 (применимо для ~42% пациентов с DMD),
- первый экзон представляет собой экзон 40, и второй экзон представляет собой экзон 53 (применимо для ~38% пациентов с DMD),
- первый экзон представляет собой экзон 44, и второй экзон представляет собой экзон 56 (применимо для ~40% пациентов с DMD),
- первый экзон представляет собой экзон 45, и второй экзон представляет собой экзон 51 (применимо для ~17% пациентов с DMD)
- первый экзон представляет собой экзон 45 и второй экзон представляет собой экзон 53 (применимо для ~28% пациентов с DMD),
- первый экзон представляет собой экзон 45, и второй экзон представляет собой экзон 55 (применимо для ~33% пациентов с DMD),
- первый экзон представляет собой экзон 45, и второй экзон представляет собой экзон 60 (применимо для ~37% пациентов с DMD), или
- первый экзон представляет собой экзон 56, и второй экзон представляет собой экзон 60 (применимо для ~2% пациентов с DMD).
Таким образом, в одном варианте осуществления олигонуклеотид по изобретению индуцирует пропускание следующих экзонов дистрофина: экзоны с 8 по 19, экзоны с 9 по 22, экзоны с 9 по 30, экзоны с 10 по 18, экзоны с 10 по 30, экзоны с 10 по 42, экзоны с 10 по 47, экзоны с 10 по 57, экзоны с 10 по 60, экзоны с 11 по 23, экзоны с 13 по 30, экзоны с 23 по 42, экзоны с 34 по 53, экзоны с 40 по 53, экзоны с 44 по 56, экзоны с 45 по 51, экзоны с 45 по 53, экзоны с 45 по 55, экзоны с 45 по 60 или экзоны с 56 по 60.
Предпочтительно, олигонуклеотид по изобретению содержит или состоит из последовательности, которая способна связываться с, нацеливаться на, гибридизоваться с и/или является обратно комплементарной области первого экзона пре-мРНК дистрофина, так что обратно комплементарная часть составляет по меньшей мере 30% длины указанного олигонуклеотида по изобретению, более предпочтительно по меньшей мере 40%, еще более предпочтительно по меньшей мере 50%, еще более предпочтительно по меньшей мере 60%, еще более предпочтительно по меньшей мере 70%, еще более предпочтительно по меньшей мере 80%, еще более предпочтительно по меньшей мере 90% или еще более предпочтительно по меньшей мере 95%, или еще более предпочтительно 98% и наиболее предпочтительно вплоть до 100%. В этом контексте первый экзон предпочтительно представляет собой экзон 8, 9, 10, 11, 13, 23, 34, 40, 44, 45 или 56 пре-мРНК дистрофина, как определено в настоящем описании. Указанный олигонуклеотид может содержать дополнительные фланкирующие последовательности. В более предпочтительном варианте осуществления длина указанной обратно комплементарной части указанного олигонуклеотида составляет по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39 или 40 нуклеотидов. Можно использовать несколько типов фланкирующих последовательностей. Предпочтительно, фланкирующие последовательности используют для модификации связывания белка с указанным олигонуклеотидом или для модификации термодинамического свойства указанного олигонуклеотида, более предпочтительно для модификации аффинности связывания РНК-мишени. В другом предпочтительном варианте осуществления дополнительные фланкирующие последовательности являются обратно комплементарными последовательностям пре-мРНК дистрофина, которые не присутствуют в указанном экзоне.
В предпочтительном варианте осуществления олигонуклеотид содержит или состоит из последовательности, которая способна связываться с, нацеливаться на, гибридизоваться с и является обратно комплементарной по меньшей мере области первого и области второго экзона дистрофина, присутствующей в пре-мРНК дистрофина, где указанные первый и второй экзоны выбраны из группы экзонов 8 (SEQ ID NO: 2), 9 (SEQ ID NO: 3), 10 (SEQ ID NO: 4), 11 (SEQ ID NO: 1761), 13 (SEQ ID NO: 1762),18 (SEQ ID NO: 5), 19 (SEQ ID NO: 6), 22 (SEQ ID NO: 7), 23 (SEQ ID NO: 8), 30 (SEQ ID NO: 9), 34 (SEQ ID NO: 1763), 40 (SEQ ID NO: 1764), 42 (SEQ ID NO: 11), 44 (SEQ ID NO: 1765), 45 (SEQ ID NO: 12), 47 (SEQ ID NO: 10), 51 (SEQ ID NO: 1760), 53 (SEQ ID NO: 13), 55 (SEQ ID NO: 14), 56 (SEQ ID NO: 15), 57 (SEQ ID NO: 1744) или 60 (SEQ ID NO: 16) и указанные области имеют по меньшей мере 10 нуклеотидов. Однако указанные области также могут иметь по меньшей мере 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200 или более нуклеотидов. Для предпочтительных экзонов, указанных выше, квалифицированный специалист способен идентифицировать область первого экзона и область второго экзона с использованием способов, известных в данной области. Более предпочтительно используют сетевой инструмент EMBOSS Matcher, как пояснено выше. Еще более предпочтительно, предпочтительные области идентичности первого и второго экзонов дистрофина, с которыми олигонуклеотид по изобретению предпочтительно связывается и/или для которых он является, по меньшей мере частично, обратно комплементарным, представлены в таблице 2. Обратная комплементарность в этом контексте предпочтительно составляет по меньшей мере 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% или 100%. Предпочтительная олигонуклеотидная последовательность, подлежащая применению в рамках изобретения, способна связываться с, гибридизоваться с, нацеливаться на и/или является обратно комплементарной области идентичности между указанными первым и вторым экзонами, предпочтительно из таблицы 2, и имеет длину по меньшей мере 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39 или 40 нуклеотидов, более предпочтительно менее 40 нуклеотидов или более предпочтительно менее 30 нуклеотидов, еще более предпочтительно менее 25 нуклеотидов и наиболее предпочтительно от 20 до 25 нуклеотидов. Обратная комплементарность в этом контексте предпочтительно составляет по меньшей мере 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% или 100%.
В одном варианте осуществления предпочтительный олигонуклеотид является таким, что первый и второй экзоны дистрофина являются такими, как указано в таблице 1, 2 или 6, и указанный олигонуклеотид способен связываться с соответствующими областями первого и второго экзонов, как указано в таблице 2 или 6 и как определяется SEQ ID NO: 17-1670, 1742, 1743 или 1766-1777.
Предпочтительные олигонуклеотиды описаны в таблице 3 и содержат или состоят из SEQ ID NO: 1671-1741. Другие предпочтительные олигонуклеотиды содержат или состоят из SEQ ID NO: 1778-1891, как описано в таблице 6. Предпочтительные олигонуклеотиды содержат SEQ ID NO: 1671-1741 или 1778-1891 и имеют на 1, 2, 3, 4 или 5 нуклеотидов больше или на 1, 2, 3, 4 или 5 нуклеотидов меньше, чем их точный SEQ ID NO, как указано в таблице 3 или 6. Эти дополнительные нуклеотиды могут присутствовать на 5'- или 3'-стороне данного SEQ ID NO. Эти отсутствующие нуклеотиды могут представлять собой нуклеотиды, присутствующие на 5'- или 3'-стороне данного SEQ ID NO. Каждый из этих олигонуклеотидов может иметь любую химическую структуру, как определено в настоящем описании ранее или их комбинации. В каждом из олигонуклеотидов, указанных в настоящем описании с помощью SEQ ID NO, U может быть заменен на T.
Более предпочтительные олигонуклеотиды приведены ниже.
Если первый экзон дистрофина представляет собой экзон 8 и второй экзон дистрофина представляет собой экзон 19, предпочтительная область экзона 8 содержит или состоит из SEQ ID NO: 17, и предпочтительная область экзона 19 содержит или состоит из SEQ ID NO: 18. Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1722 или 1723 или содержит SEQ ID NO: 1722 или 1723, и имеет длину 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 13, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 101, и предпочтительная область экзона 13 содержит или состоит из SEQ ID NO: 102.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 14, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 103, и предпочтительная область экзона 14 содержит или состоит из SEQ ID NO: 104.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 15, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 105, и предпочтительная область экзона 15 содержит или состоит из SEQ ID NO: 106.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 18, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 109, и предпочтительная область экзона 18 содержит или состоит из SEQ ID NO: 110. Предпочтительные олигонуклеотиды содержат:
- последовательность оснований, определяемую любой из SEQ ID NO: 1679-1681, 1778, 1812, 1813, 1884-1886, 1890 или 1891 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую SEQ ID NO: 1814 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1815-1819 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1820, 1824 и имеющую длину 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1826, 1782, 1832 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1821, 1825, 1780 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1822 и имеющую длину 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1823, 1781, 1829, 1830, 1831 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов,
- последовательность оснований, определяемую любой из SEQ ID NO: 1887 и имеющую длину 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1888 или 1889 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую SEQ ID NO: 1827 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую SEQ ID NO: 1828 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 18, другая предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 1766, и другая предпочтительная область экзона 18 содержит или состоит из SEQ ID NO: 1767. Предпочтительные олигонуклеотиды содержат последовательность оснований, определяемую любой из SEQ ID NO: 1783, 1833, 1834, 1835 и имеющую длину 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 18, другая предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 1768, и другая предпочтительная область экзона 18 содержит или состоит из SEQ ID NO: 1769. Предпочтительные олигонуклеотиды содержат последовательность оснований, определяемую любой из SEQ ID NO: 1673 или 1674 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 20, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 111, и предпочтительная область экзона 20 содержит или состоит из SEQ ID NO: 112.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 27, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 121, и предпочтительная область экзона 27 содержит или состоит из SEQ ID NO: 122.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 30, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 127, и предпочтительная область экзона 30 содержит или состоит из SEQ ID NO: 128. Предпочтительные олигонуклеотиды содержат:
- последовательность оснований, определяемую любой из SEQ ID NO: 1679-1681, 1812, 1813, 1884-1886, 1890 или 1891 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую SEQ ID NO: 1814 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую SEQ ID NO: 1675 или 1676 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую SEQ ID NO: 1677 или 1678 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1784, 1836 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1786, 1838 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1780 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1785, 1837 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 30, другая предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 1772, и другая предпочтительная область экзона 30 содержит или состоит из SEQ ID NO: 1773. Предпочтительные олигонуклеотиды содержат:
- последовательность оснований, определяемую любой из SEQ ID NO: 1688, 1689, 1839, 1840, 1841, 1842, 1843 или 1844 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1845, 1846, 1847, 1848 и имеющую длину 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1849, 1850 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1787, 1851 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 31, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 129, и предпочтительная область экзона 31 содержит или состоит из SEQ ID NO: 130.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 32, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 131, и предпочтительная область экзона 32 содержит или состоит из SEQ ID NO: 132.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 35, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 137, и предпочтительная область экзона 35 содержит или состоит из SEQ ID NO: 138.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 42, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 151, и предпочтительная область экзона 42 содержит или состоит из SEQ ID NO: 152.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 44, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 153, и предпочтительная область экзона 44 содержит или состоит из SEQ ID NO: 154.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 47, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 157, и предпочтительная область экзона 47 содержит или состоит из SEQ ID NO: 158.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 48, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 159, и предпочтительная область экзона 48 содержит или состоит из SEQ ID NO: 160.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 55, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 167, и предпочтительная область экзона 55 содержит или состоит из SEQ ID NO: 168.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 57, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 169, и предпочтительная область экзона 57 содержит или состоит из SEQ ID NO: 170.
Если первый экзон дистрофина представляет собой экзон 10 и второй экзон дистрофина представляет собой экзон 60, предпочтительная область экзона 10 содержит или состоит из SEQ ID NO: 173, и предпочтительная область экзона 60 содержит или состоит из SEQ ID NO: 174.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1673 или содержит SEQ ID NO: 1673 и имеет длину 25, 26, 27, 28, 29 или 30 нуклеотидов.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1675 или содержит SEQ ID NO: 1675 и имеет длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1677 или содержит SEQ ID NO: 1677 и имеет длину 25, 26, 27, 28, 29 или 30 нуклеотидов.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1679 или содержит SEQ ID NO: 1679 и имеет длину 25, 26, 27, 28, 29 или 30 нуклеотидов. Предпочтительный олигонуклеотид содержит последовательность оснований SEQ ID NO: 1679 и имеет длину 25, 26, 27, 28, 29 или 30 нуклеотидов. Необязательно, 1, 2, 3, 4, 5, 6, 7 из U в SEQ ID NO: 1679 заменены на T. В предпочтительном варианте осуществления все U в SEQ ID NO: 1679 заменен/заменены на T. Предпочтительный олигонуклеотид, содержащий SEQ ID NO: 1679, содержит любую из химических структур, определенных в настоящем описании выше: модификацию основания и/или модификацию сахара и/или модификацию основной цепи.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1681 или содержит SEQ ID NO: 1681 и имеет длину 25, 26, 27, 28, 29 или 30 нуклеотидов.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1684 или содержит SEQ ID NO: 1684 и имеет длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1685 или содержит SEQ ID NO: 1685 и имеет длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1686 или содержит SEQ ID NO: 1686 и имеет длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1688 или содержит SEQ ID NO: 1688 и имеет длину 26, 27, 28, 29 или 30 нуклеотидов. Предпочтительный олигонуклеотид содержит последовательность оснований SEQ ID NO: 1688 и имеет длину 26, 27, 28, 29 или 30 нуклеотидов. Необязательно, 1, 2, 3, 4, 5, 6 из U в SEQ ID NO: 1688 заменен/заменены на T. В предпочтительном варианте осуществления все U в SEQ ID NO: 1688 заменены на T. Предпочтительный олигонуклеотид, содержащий SEQ ID NO: 1688, содержит любую из химических структур, определенных в настоящем описании выше: модификацию основания и/или модификацию сахара и/или модификацию основной цепи.
Для каждого из олигонуклеотидов, соответствующих SEQ ID NO: 1673, 1675, 1677, 1679, 1681, 1684, 1685, 1686 и 1688, первый экзон дистрофина представляет собой экзон 10. Однако второй экзон дистрофина представляет собой экзон 13, 14, 15, 18, 20, 27, 30, 31, 32, 35, 42, 44, 47, 48, 55, 57 или 60. Это означает, что при использовании любого из этих олигонуклеотидов может произойти образование нескольких транскриптов в рамке считывания, каждый из которых приводит к продукции укороченного, но (полу)функционального белка дистрофина.
Если первый экзон дистрофина представляет собой экзон 11 и второй экзон дистрофина представляет собой экзон 23, предпочтительная область экзона 23 содержит или состоит из SEQ ID NO: 191, и предпочтительная область экзона 23 содержит или состоит из SEQ ID NO: 192. Предпочтительные олигонуклеотиды содержат:
- последовательность оснований, определяемую любой из SEQ ID NO: 1794, 1861, 1795, 1862 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1796, 1863, 1797, 1864 и имеющую длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1798, 1865, 1799, 1866 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 13 и второй экзон дистрофина представляет собой экзон 30, предпочтительная область экзона 13 содержит или состоит из SEQ ID NO: 285, и предпочтительная область экзона 30 содержит или состоит из SEQ ID NO: 286. Предпочтительные олигонуклеотиды содержат:
- последовательность оснований, определяемую любой из SEQ ID NO: 1808, 1867, 1809, 1868, 1810, 1869, 1858, 1873 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов,
- последовательность оснований, определяемую любой из SEQ ID NO: 1811, 1870, 1859, 1874 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1856, 1871, 1860, 1875 и имеющую длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1857, 1872 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 23 и второй экзон дистрофина представляет собой экзон 42, предпочтительная область экзона 23 содержит или состоит из SEQ ID NO: 776, и предпочтительная область экзона 42 содержит или состоит из SEQ ID NO: 777. Предпочтительные олигонуклеотиды содержат или состоят из SEQ ID NO: 1698-1703.
Если первый экзон дистрофина представляет собой экзон 34 и второй экзон дистрофина представляет собой экзон 53, предпочтительная область экзона 34 содержит или состоит из SEQ ID NO: 1294, и предпочтительная область экзона 53 содержит или состоит из SEQ ID NO: 1295. Предпочтительные олигонуклеотиды содержат:
- последовательность оснований, определяемую любой из SEQ ID NO: 1800, 1876, 1801, 1877 и имеющую длину 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1802, 1878, 1803, 1879 и имеющую длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 40 и второй экзон дистрофина представляет собой экзон 53, предпочтительная область экзона 40 содержит или состоит из SEQ ID NO: 1477, и предпочтительная область экзона 53 содержит или состоит из SEQ ID NO: 1478. Предпочтительные олигонуклеотиды содержат:
- последовательность оснований, определяемую любой из SEQ ID NO: 1804, 1880 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
- последовательность оснований, определяемую любой из SEQ ID NO: 1805, 1881 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 44 и второй экзон дистрофина представляет собой экзон 56, предпочтительная область экзона 44 содержит или состоит из SEQ ID NO: 1577, и предпочтительная область экзона 56 содержит или состоит из SEQ ID NO: 1558. Предпочтительные олигонуклеотиды содержат последовательность оснований, определяемую любой из SEQ ID NO: 1806, 1882, 1807, 1883 и имеющую длину 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 45 и второй экзон дистрофина представляет собой экзон 51, предпочтительная область экзона 45 содержит или состоит из SEQ ID NO: 1567, и предпочтительная область экзона 51 содержит или состоит из SEQ ID NO: 1568. Предпочтительные олигонуклеотиды содержат или состоят из SEQ ID NO: 1730-1731.
Если первый экзон дистрофина представляет собой экзон 45 и второй экзон дистрофина представляет собой экзон 53, предпочтительная область экзона 45 содержит или состоит из SEQ ID NO: 1569, и предпочтительная область экзона 53 содержит или состоит из SEQ ID NO: 1570. Предпочтительные олигонуклеотиды содержат или состоят из SEQ ID NO: 1732-1737.
Если первый экзон дистрофина представляет собой экзон 45 и второй экзон дистрофина представляет собой экзон 55, предпочтительная область экзона 45 содержит или состоит из SEQ ID NO: 1571, и предпочтительная область экзона 55 содержит или состоит из SEQ ID NO: 1572. Предпочтительные олигонуклеотиды содержат или состоят из SEQ ID NO: 1704-1719, 1788, 1852, 1789, 1853.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1706 или содержит SEQ ID NO: 1706 и имеет длину 25, 26, 27, 28, 29 или 30 нуклеотидов. Предпочтительный олигонуклеотид, содержащий SEQ ID NO: 1706 и имеющий длину 25 нуклеотидов, состоит из SEQ ID NO: 1706.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1707 или содержит SEQ ID NO: 1707 и имеет длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов. Предпочтительный олигонуклеотид, содержащий SEQ ID NO: 1707 и имеющий длину 25 нуклеотидов, состоит из SEQ ID NO: 1706.
Предпочтительный олигонуклеотид состоит из SEQ ID NO: 1713 или содержит SEQ ID NO: 1713 и имеет длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов. Предпочтительный олигонуклеотид, содержащий SEQ ID NO: 1713 и имеющий длину 25 нуклеотидов, состоит из SEQ ID NO: 1710.
Предпочтительные олигонуклеотиды содержат последовательность оснований, определяемую любой из SEQ ID NO: 1788, 1852, 1789, 1853 и имеющую длину 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 45 и второй экзон дистрофина представляет собой экзон 55, другая предпочтительная область экзона 45 содержит или состоит из SEQ ID NO: 1774, и другая предпочтительная область экзона 55 содержит или состоит из SEQ ID NO: 1775. Предпочтительные олигонуклеотиды содержат последовательность оснований, определяемую любой из SEQ ID NO: 1790, 1854, 1792, 1855 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов.
Если первый экзон дистрофина представляет собой экзон 45 и второй экзон дистрофина представляет собой экзон 60, предпочтительная область экзона 45 содержит или состоит из SEQ ID NO: 1577, и предпочтительная область экзона 60 содержит или состоит из SEQ ID NO: 1578. Предпочтительные олигонуклеотиды содержат или состоят из SEQ ID NO: 1738-1741.
Если первый экзон дистрофина представляет собой экзон 56 и второй экзон дистрофина представляет собой экзон 60, предпочтительная область экзона 56 содержит или состоит из SEQ ID NO: 1742, и предпочтительная область экзона 60 содержит или состоит из SEQ ID NO: 1743. Предпочтительные олигонуклеотиды содержат или состоят из SEQ ID NO: 1720-1721.
Более предпочтительный олигонуклеотид содержит:
(a) последовательность оснований, определяемую SEQ ID NO: 1673 или 1674 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов; или
(b) последовательность оснований, определяемую SEQ ID NO: 1675 или 1676 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов; или
(c) последовательность оснований, определяемую SEQ ID NO: 1677 или 1678 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов; или
(d) последовательность оснований, определяемую любой из SEQ ID NO: 1679-1681 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов; или
(e) последовательность оснований, определяемую любой из SEQ ID NO: 1684-1686 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов; или
(f) последовательность оснований, определяемую SEQ ID NO: 1688 или 1689 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов; или
(g) последовательность оснований, определяемую любой из SEQ ID NO: 1704-1706 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов,
(h) последовательность оснований, определяемую любой из SEQ ID NO: 1707-1709 и имеющую длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов; или
(i) последовательность оснований, определяемую любой из SEQ ID NO: 1710, 1713-1717 и имеющую длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Еще более предпочтительные олигонуклеотиды содержат:
(a) последовательность оснований, определяемую любой из SEQ ID NO: 1679-1681, 1778, 1812, 1813, 1884-1886, 1890 или 1891 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или SEQ ID NO: 1814 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов, или
(b) последовательность оснований, определяемую SEQ ID NO: 1688, 1689 или 1839-1844 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов, или
(c) последовательность оснований, определяемую SEQ ID NO: 1673 или 1674 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(d) последовательность оснований, определяемую SEQ ID NO: 1675 или 1676 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(e) последовательность оснований, определяемую SEQ ID NO: 1677 или 1678 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(f) последовательность оснований, определяемую любой из SEQ ID NO: 1684-1686 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(g) последовательность оснований, определяемую любой из SEQ ID NO: 1704-1706 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(h) последовательность оснований, определяемую любой из SEQ ID NO: 1707-1709 и имеющую длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(i) последовательность оснований, определяемую любой из SEQ ID NO: 1710, 1713-1717 и имеющую длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(j) последовательность оснований, определяемую любой из SEQ ID NO: 1815-1819 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(k) последовательность оснований, определяемую любой из SEQ ID NO: 1820, 1824, и имеющую длину 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(l) последовательность оснований, определяемую любой из SEQ ID NO: 1826, 1782, 1832 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(m) последовательность оснований, определяемую любой из SEQ ID NO: 1821, 1825, 1780 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(n) последовательность оснований, определяемую любой из SEQ ID NO: 1822 и имеющую длину 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(o) последовательность оснований, определяемую любой из SEQ ID NO: 1823, 1781, 1829, 1830, 1831 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(p) последовательность оснований, определяемую любой из SEQ ID NO: 1783, 1833, 1834, 1835 и имеющую длину 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(q) последовательность оснований, определяемую любой из SEQ ID NO: 1887 и имеющую длину 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(r) последовательность оснований, определяемую любой из SEQ ID NO: 1888 или 1889 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов, или
(s) последовательность оснований, определяемую SEQ ID NO: 1827 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(t) последовательность оснований, определяемую SEQ ID NO: 1828 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(u) последовательность оснований, определяемую любой из SEQ ID NO: 1784, 1836 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(v) или последовательность оснований, определяемую любой из SEQ ID NO: 1786, 1838 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(w) последовательность оснований, определяемую любой из SEQ ID NO: 1780 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(x) последовательность оснований, определяемую любой из SEQ ID NO: 1785, 1837 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(y) последовательность оснований, определяемую любой из SEQ ID NO: 1845, 1846, 1847, 1848 и имеющую длину 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(z) последовательность оснований, определяемую любой из SEQ ID NO: 1849, 1850 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(a1) последовательность оснований, определяемую любой из SEQ ID NO: 1787, 1851 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов, или
(b1) последовательность оснований, определяемую любой из SEQ ID NO: 1788, 1852, 1789, 1853 и имеющую длину 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(c1) последовательность оснований, определяемую любой из SEQ ID NO: 1790, 1854, 1792, 1855 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(d1) последовательность оснований, определяемую любой из SEQ ID NO: 1794, 1861, 1795, 1862 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(e1) последовательность оснований, определяемую любой из SEQ ID NO: 1796, 1863, 1797, 1864 и имеющую длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(f1) последовательность оснований, определяемую любой из SEQ ID NO: 1798, 1865, 1799, 1866 и имеющую длину 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(g1) последовательность оснований, определяемую любой из SEQ ID NO: 1808, 1867, 1809, 1868, 1810, 1869, 1858, 1873 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(h1) последовательность оснований, определяемую любой из SEQ ID NO: 1811, 1870, 1859, 1874 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(i1) последовательность оснований, определяемую любой из SEQ ID NO: 1856, 1871, 1860, 1875 и имеющую длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(j1) последовательность оснований, определяемую любой из SEQ ID NO: 1857, 1872 и имеющую длину 26, 27, 28, 29 или 30 нуклеотидов, или
(k1) последовательность оснований, определяемую любой из SEQ ID NO: 1800, 1876, 1801, 1877 и имеющую длину 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(l1) последовательность оснований, определяемую любой из SEQ ID NO: 1802, 1878, 1803, 1879 и имеющую длину 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(m1) последовательность оснований, определяемую любой из SEQ ID NO: 1804, 1880 и имеющую длину 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(n1) последовательность оснований, определяемую любой из SEQ ID NO: 1805, 1881 и имеющую длину 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, или
(o1) последовательность оснований, определяемую любой из SEQ ID NO: 1806, 1882, 1807, 1883 и имеющую длину 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов.
Олигонуклеотид по изобретению, который содержит или состоит из последовательности, определяемой номером SEQ ID, также охватывает олигонуклеотид, содержащий последовательность оснований, определяемую этим SEQ ID. Олигонуклеотиды, имеющие модифицированную основную цепь (т.е. модифицированные части сахаров и/или модифицированные межнуклеозидные связи), относительно олигонуклеотидов, определяемых SEQ ID, также охватываются изобретением. Каждое основание U в SEQ ID NO олигонуклеотида, как определено в настоящем описании, может быть модифицировано или заменено на T.
Композиция
В следующем аспекте предусматривается композиция, содержащая олигонуклеотид, как описано в предыдущем разделе под названием "Олигонуклеотид". Эта композиция предпочтительно содержит или состоит из олигонуклеотида, как описано выше. Предпочтительная композиция содержит один единственный олигонуклеотид, как определено выше. Таким образом, понятно, что пропускания по меньшей мере указанного первого и указанного второго экзона достигают с использованием одного единственного олигонуклеотида, а не с использованием коктейля различных олигонуклеотидов.
В предпочтительном варианте осуществления указанная композиция предназначена для применения в качестве лекарственного средства. Таким образом, указанная композиция представляет собой фармацевтическую композицию. Фармацевтическая композиция обычно содержит фармацевтически приемлемый носитель, разбавитель и/или эксципиент. В предпочтительном варианте осуществления композиция по настоящему изобретению содержит соединение, как определено в настоящем описании, и необязательно дополнительно содержит фармацевтически приемлемый состав, наполнитель, консервант, солюбилизатор, носитель, разбавитель, эксципиент, соль, адъювант и/или растворитель. Такой фармацевтически приемлемый носитель, наполнитель, консервант, солюбилизатор, разбавитель, соль, адъювант, растворитель и/или эксципиент может быть найден, например, в Remington. Соединение, как описано в рамках изобретения, обладает по меньшей мере одной ионогенной группой. Ионогенная группа может представлять собой основание или кислоту, и она может быть заряженной или нейтральной. Ионогенная группа может присутствовать в качестве пары ионов с соответствующим противоионом, который имеет противоположный заряд(ы). Примерами катионных противоионов являются натрий, калий, цезий, Tris, литий, кальций, магний, триалкиламмоний, триэтиламмоний и тетраалкиламмоний. Примерами анионных противоионов являются хлорид, бромид, йодид, лактат, мезилат, ацетат, трифторацетат, дихлорацетат и цитрат. Примеры противоионов описаны (Kumar L., которая включена в настоящее описание в качестве ссылки в полном объеме). Таким образом, в предпочтительном варианте осуществления олигонуклеотид по изобретению приводят в контакт с композицией, содержащей такие ионогенные группы, предпочтительно Ca2+, с получением хелатного комплекса олигонуклеотида, содержащего два или более идентичных олигонуклеотидов, связанных таким мультивалентным катионом, как уже определено в настоящем описании.
Фармацевтическую композицию, кроме того, можно составлять, чтобы дополнительно способствовать повышению стабильности, растворимости, всасывания, биодоступности, фармакокинетики и клеточного захвата указанного соединения, в частности, составов, содержащих эксципиенты или конъюгаты, способные образовывать комплексы, наночастицы, микрочастицы, нанотрубки, наногели, гидрогели, полоксамеры или плюроники, полимерсомы, коллоиды, микропузырьки, везикулы, мицеллы, липоплексы и/или липосомы. Примеры наночастиц включают полимерные наночастицы, золотые наночастицы, магнитные наночастицы, частицы на основе диоксида кремния, липидные наночастицы, наночастицы на основе сахаров, белковые наночастицы и пептидные наночастицы.
Предпочтительная композиция содержит по меньшей мере один эксципиент, который может далее способствовать усилению нацеливания и/или доставки указанной композиции и/или указанного олигонуклеотида к и/или в мышцу и/или клетку. Клетка может представлять собой мышечную клетку.
Другая предпочтительная композиция может содержать по меньшей мере один эксципиент, классифицируемый как второй тип эксципиента. Второй тип эксципиента может включать или содержать конъюгатную группу, как описано в настоящем описании, для усиления нацеливания и/или доставки композиции и/или олигонуклеотида по изобретению к ткани и/или клетке и/или в ткань и/или клетку, например, такую как мышечная ткань или клетка. Оба типа эксципиентов можно комбинировать в одну единственную композицию, как определено в настоящем описании. Предпочтительные конъюгатные группы описаны в части, посвященной определениям.
Квалифицированный специалист может выбирать, комбинировать и/или адаптировать один или несколько из описанных выше или других альтернативных эксципиентов и систем для доставки для составления и доставки соединения для применения в рамках настоящего изобретения.
Такую фармацевтическую композицию по изобретению можно вводить в эффективной концентрации в установленные моменты времени животному, предпочтительно млекопитающему. Более предпочтительным млекопитающим является человек. Соединение или композиция, как определено в настоящем описании, могут быть пригодны для прямого введения в клетку, ткань и/или орган in vivo у индивидуумов, предпочтительно указанные индивидуумы страдают или имеют риск развития BMD или DMD, и их можно вводить прямо in vivo, ex vivo или in vitro. Введение можно осуществлять системным и/или парентеральным путями, например, внутривенным, подкожным, внутрижелудочковым, интратекальным, внутримышечным, интраназальным, энтеральным, интравитреальным, внутрицеребральным, эпидуральным или пероральным путем.
Предпочтительно, такая фармацевтическая композиция по изобретению может быть инкапсулирована в форме эмульсии, суспензии, пилюли, таблетки, капсулы или мягкой желатиновой капсулы для пероральной доставки, или она может быть в форме аэрозоля или сухого порошка для доставки в дыхательные пути и легкие.
В одном варианте осуществления соединение по изобретению можно использовать вместе с другим соединением, уже известным тем, что его используют для лечения указанного заболевания. Такие другие соединения можно использовать для замедления прогрессирования заболевания, для снижения аномального поведения или движений, для снижения воспаления мышечной ткани, для улучшения функции, целостности и/или выживаемости мышечных волокон и/или для улучшения, увеличения или восстановления сердечной функции. Примерами являются, но не ограничиваясь ими, стероид, предпочтительно (глюко)кортикостероид, ингибитор ACE (предпочтительно периндоприл), блокатор рецептора ангиотензина II типа 1 (предпочтительно лозартан), ингибитор фактора некроза опухоли-альфа (TNFα), ингибитор TGFβ (предпочтительно декорин), рекомбинантный бигликан человека, источник mIGF-1, ингибитор миостатина, манноза-6-фосфат, антиоксидант, ингибитор ионных каналов, ингибитор протеаз, ингибитор фосфодиэстеразы (предпочтительно ингибитор PDE5, такой как силденафил или тадалафил), L-аргинин, блокаторы дофамина, амантадин, тетрабеназин и/или кофермент Q10. Это комбинированное применение может представлять собой последовательное применение: каждый компонент вводят в отдельной композиции. Альтернативно, каждое соединение можно использовать вместе в одной композиции.
Применение
В следующем аспекте предусматривается применение композиции или соединения, как описано в настоящем описании, в качестве лекарственного средства или части терапии, или применения, в которых соединение проявляет его активность внутриклеточно.
В предпочтительном варианте осуществления соединение или композиция по изобретению предназначены для применения в качестве лекарственного средства, где лекарственное средство предназначено для профилактики, замедления, смягчения и/или лечения заболевания, как определено в настоящем описании, предпочтительно DMD или BMD.
Способ профилактики, замедления, смягчения и/или лечения заболевания
В следующем аспекте предусматривается способ профилактики, замедления, смягчения и/или лечения заболевания, как определено в настоящем описании, предпочтительно DMD или BMD. Указанное заболевание можно предупреждать, лечить, замедлять или смягчать у индивидуума, в клетке, ткани или органе указанного индивидуума. Способ включает введение олигонуклеотида или композиции по изобретению указанному индивидууму или субъекту, нуждающемуся в этом.
Способ по изобретению, где олигонуклеотид или композиция, как определено в настоящем описании, могут быть пригодными для введения в клетку, ткань и/или орган in vivo индивидуумам, предпочтительно индивидуумам, страдающим BMD или DMD или имеющим риск развития такого заболевания, можно проводить in vivo, ex vivo или in vitro. Индивидуум или субъект, нуждающиеся в этом, предпочтительно представляют собой млекопитающее, более предпочтительно человека.
В одном варианте осуществления в способе по изобретению концентрация олигонуклеотида или композиции находится в диапазоне от 0,01 нМ до 1 мкМ. Более предпочтительно, используемая концентрация составляет от 0,02 до 400 нМ, или от 0,05 до 400 нМ, или от 0,1 до 400 нМ, еще более предпочтительно от 0,1 до 200 нМ.
Диапазоны доз олигонуклеотида или композиции по изобретению предпочтительно выбирают, исходя из исследований с возрастающей дозой в клинических испытаниях (применение in vivo), для которых существуют строгие требования протокола. Олигонуклеотид, как определено в настоящем описании, можно использовать в дозе, которая находится в диапазоне от 0,01 до 200 мг/кг, или от 0,05 до 100 мг/кг, или от 0,1 до 50 мг/кг, или от 0,1 до 20 мг/кг, предпочтительно от 0,5 до 10 мг/кг.
Диапазоны концентрации или дозы олигонуклеотида или композиции, приведенные выше, являются предпочтительными концентрациями или дозами для применений in vitro или ex vivo. Квалифицированному специалисту будет понятно, что в зависимости от типа используемого олигонуклеотида, клетки-мишени, подлежащей лечению, гена-мишени и его уровней экспрессии, используемой среды и условий трансфекции и инкубации, концентрация или доза указанного используемого олигонуклеотида может далее варьировать и может требовать дальнейшей оптимизации.
Определения
"Идентичность последовательностей", как известно в данной области, представляет собой взаимосвязь между двумя или более последовательностями нуклеиновой кислоты (полинуклеотид или нуклеотид), как определяют путем сравнения последовательностей. В данной области процент "идентичности" или "сходства" указывает на степень родственности последовательностей нуклеиновой кислоты при определении по соответствию цепей таких последовательностей. "Идентичность" может быть заменена в настоящем описании "сходством". Предпочтительно, процент идентичности определяют путем сравнения всей SEQ ID NO, как определено в настоящем описании. Однако также можно использовать часть последовательности. Часть последовательности в этом контексте может означать по меньшей мере 5%, 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или 100% данной последовательности или SEQ ID NO.
"Идентичность" и "сходство" можно без труда вычислить известными способами, включая, но не ограничиваясь ими, способы, описанные в Computational Molecular Biology, Lesk, A.M., ed., Oxford University Press, New York, 1988; Biocomputing: Informatics and Genome Projects, Smith, D.W., ed., Academic Press, New York, 1993; Computer Analysis of Sequence Data, Part I, Griffin, A.M., and Griffin, H.G., eds., Humana Press, New Jersey, 1994; Sequence Analysis in Molecular Biology, von Heine, G., Academic Press, 1987; и Sequence Analysis Primer Gribskov, M. and Devereux, J., eds., M Stockton Press, New York, 1991; и Carillo, H., and Lipman, D., SIAM J. Applied Math., 48: 1073 (1988).
Предпочтительные способы определения идентичности разработаны так, чтобы обеспечить наибольшее соответствие между исследуемыми последовательностями. Способы определения идентичности и сходства закодированы в общедоступных компьютерных программах. Предпочтительные компьютерные программы для определения идентичности и сходства между двумя последовательностями включают, например, пакет программ GCG (Devereux, J., et al., Nucleic Acids Research 12 (1):387 (1984)), BestFit и FASTA (Altschul, S.F. et al., J. Mol. Biol. 215: 403-410 (1990)). Семейство программ BLAST 2.0, которое можно использовать для поиска сходства в базах данных, включает, например, BLASTN для нуклеотидных последовательностей запроса против нуклеотидных последовательностей из баз данных. Семейство программ BLAST 2.0 является общедоступным от NCBI и других источников (BLAST Manual, Altschul, S., et al., NCBI NLM NIH Bethesda, MD 20894; Altschul, S., et al., J. Mol. Biol. 215: 403-410 (1990)). Также для определения идентичности можно использовать хорошо известный алгоритм Смита-Ватермана.
Предпочтительные параметры для сравнения нуклеиновых кислот включают следующие параметры: алгоритм: Needleman and Wunsch, J. Mol. Biol. 48: 443-453 (1970); матрица сравнений: совпадение = +10, несовпадение = 0; штраф за пропуск: 50; штраф за продолжение пропуска: 3. Доступен в качестве программы Gap от Genetics Computer Group, расположенной в Madison, Wis. Выше приведены параметры по умолчанию для сравнений нуклеиновых кислот.
Другим предпочтительным способом определения сходства и идентичности последовательностей является способ с использованием алгоритма Needleman-Wunsch (Needleman, S.B. and Wunsch, C.D. (1970) J. Mol. Biol. 48, 443-453, Kruskal, J.B. (1983), обзор сравнения последовательностей: D. Sankoff and J.B. Kruskal, (ed.), Time warps, string edits and macromolecules: the theory and practice of sequence comparison, pp. 1-44 Addison Wesley).
Другим предпочтительным способом определения сходства и идентичности последовательностей является способ с использованием EMBOSS Matcher и алгоритма Waterman-Eggert (локальное выравнивание двух последовательностей; [Schoniger и Waterman, Bulletin of Mathematical Biology 1992, Vol. 54 (4), pp. 521-536; Vingron and Waterman, J. Mol. Biol. 1994; 235(1), p1-12]). Можно использовать следующие web-сайты: http://www.ebi.ac.uk/Tools/emboss/align/index.html или http://emboss.bioinformatics.nl/cgi-bin/emboss/matcher. Определения параметров, используемых в этом алгоритме, находится на следующем web-сайте: http://emboss.sourceforge.net/docs/themes/AlignFormats.html#id. Предпочтительно используют параметры по умолчанию (матрица: EDNAFULL, штраф за пропуск: 16, штраф за продолжение пропуска: 4). Emboss Matcher обеспечивает наилучшее локальное выравнивание между двумя последовательностями, однако также предоставляются альтернативные выравнивания. В таблице 2 представлено наилучшее локальное выравнивание между двумя различными экзонами, предпочтительно экзонами дистрофина, которые предпочтительно используют для конструирования олигонуклеотидов. Однако также можно идентифицировать альтернативные выравнивания и, таким образом, альтернативные области идентичности между двумя экзонами, и их можно использовать для конструирования олигонуклеотидов, что также является частью настоящего изобретения.
На протяжении заявки слова "связывается", "нацеливается", "гибридизуется" могут использоваться взаимозаменяемо, когда их используют в контексте олигонуклеотида, который обратно комплементарен области пре-мРНК, как определено в настоящем описании. Аналогично, выражения "способен связываться", "способен нацеливаться" и "способен гибридизоваться" могут использоваться взаимозаменяемо для указания на то, что олигонуклеотид имеет определенную последовательность, которая позволяет связывание, нацеливание или гибридизацию с последовательностью-мишенью. Когда последовательность-мишень определена в настоящем описании или известна в данной области, квалифицированный специалист способен сконструировать все возможные структуры указанного олигонуклеотида с использованием принципа обратной комплементарности. В этом отношении будет понятно, что допустимо ограниченное количество несоответствий или пропусков в последовательности между олигонуклеотидом по изобретению и последовательностями-мишенями в первом и/или втором экзонах при условии, что связывание не изменяется, как рассмотрено выше. Таким образом, олигонуклеотид, который способен связываться с определенной последовательностью-мишенью, может считаться олигонуклеотидом, который обратно комплементарен последовательности-мишени. В контексте изобретения "гибридизуется" или "способен гибридизоваться" используют для физиологических условий в клетке, предпочтительно в клетке человека, если нет иных указаний.
Как используют в рамках изобретения, "гибридизация" относится к спариванию комплементарных олигомерных соединений (например, антисмысловое соединение и его нуклеиновая кислота-мишень). Не ограничиваясь конкретным механизмом, наиболее распространенный механизм образования пар вовлекает образование водородных связей, которое может представлять собой образование водородных связей по типу Уотсона-Крика, Хугстина или обратному хугстиновскому типу между комплементарными нуклеозидными или нуклеотидными основаниями (нуклеиновые основания). Например, природное основание аденин представляет собой нуклеиновое основание, комплементарное природному нуклеиновому основанию тимину, 5-метилурацилу и урацилу, которые образуют пару путем образования водородных связей. Природное основание гуанин представляет собой нуклеиновое основание, комплементарное природным основаниям цитозину и 5-метилцитозину. Гибридизация может происходить в различных условиях.
Аналогично, "обратную комплементарность" используют для указания на две нуклеотидных последовательности, которые способны гибридизоваться друг с другом, в то время как одна из последовательностей ориентирована от 3' к 5', а другая ориентирована в обратном направлении, т.е. от 5' к 3'. Таким образом, нуклеозид A на первой нуклеотидной последовательности способен образовывать пару с нуклеозидом A* на второй нуклеотидной последовательности через их соответствующие нуклеиновые основания, и нуклеозид B, расположенный в 5'-положении от упомянутого выше нуклеозида A в первой нуклеотидной последовательности, способен образовывать пару с нуклеозидом B*, который расположен в 3'-положении от упомянутого выше нуклеозида A* во второй нуклеотидной последовательности. В контексте настоящего изобретения первая нуклеотидная последовательность, как правило, представляет собой олигонуклеотид по изобретению, и вторая нуклеотидная последовательность представляет собой часть экзона из пре-мРНК, предпочтительно пре-мРНК дистрофина. Олигонуклеотид предпочтительно называют обратно комплементарным области экзона (первого и/или второго экзона), когда указанный олигонуклеотид по меньшей мере на 45, 50, 55, 60, 65, 70, 75, 80, 85, 90, 95, 100% обратно комплементарен указанной области указанного первого и/или второго экзона. Предпочтительно, обратная комплементарность составляет по меньшей мере 80%, 85%, 90%, 95% или 100%.
В контексте изобретения эксципиенты включают полимеры (например, полиэтиленимин (PEI), полипропиленимин (PPI), производные декстрана, бутилцианоакрилат (PBCA), гексилцианоакрилат (PHCA), сополимер молочной и гликолевой кислоты (PLGA), полиамины (например, спермин, спермидин, путресцин, кадаверин), хитозан, поли(амидоамины) (PAMAM), поли(сложный эфир-амин), поливиниловый эфир, поливинилпирролидон (PVP), полиэтиленгликоль (PEG) циклодекстрины, гиалуроновую кислоту, коломиновую кислоту и их производные), дендримеры (например, поли(амидоамин)), липиды {например, 1,2-диолеоил-3-диметиламмоний пропан (DODAP), диолеоилдиметиламмоний хлорид (DODAC), производные фосфатидилхолина [например, 1,2- дистеароил-sn-глицеро-3-фосфохолин (DSPC)], производные лизо-фосфатидилхолина [например, 1-стеароил-2-лизо-sn-глицеро-3-фосфохолин (S-LysoPC)], сфингомиелин, 2-{3-[бис-(3-аминопропил)амино]пропиламино}-N-дитетрацедилкарбамоилметилацетамид (RPR209120), производные фосфоглицерина [например, 1,2-дипальмитоил-sn-глицеро-3-фосфоглицерин, натриевая соль (DPPG-Na), производные фосфатицидной кислоты [1,2-дистеароил-sn-глицеро-3-фосфатицидная кислота, натриевая соль (DSPA), производные фосфатидилэтаноламина [например, диолеоил-фосфатидилэтаноламин (DOPE), 1,2-дистеароил-sn-глицеро-3-фосфоэтаноламин (DSPE), 2-дифитаноил-sn-глицеро-3-фосфоэтаноламин (DPhyPE)], N-[1-(2,3-диолеоилокси)пропил]-N,N,N-триметиламмоний (DOTAP), N-[1-(2,3-диолеоилокси)пропил]-N,N,N-триметиламмоний (DOTMA), 1,3-диолеоилокси-2-(6-карбоксиспермил)пропиламид (DOSPER), (1,2-димиристоилоксипропил-3-диметилгидроксиэтиламмоний (DMRIE), (N1-холестерилоксикарбонил-3,7-диазанонан-1,9-диамин (CDAN), диметилдиоктадециламмоний бромид (DDAB), 1-пальмитоил-2-олеоил-sn-глицерол-3-фосфохолин (POPC), (b-L-аргинил-2,3-L-диаминопропионовая кислота-N-пальмитил-N-олеиламид тригидрохлорид (AtuFECT01), производные N,N-диметил-3-аминопропана [например, 1,2-дистеароилокси-N,N-диметил-3-аминопропан (DSDMA), 1,2-диолеилокси-N,N-диметил-3-аминопропан (DoDMA), 1,2-дилинолеилокси-N,N-3-диметиламинопропан (DLinDMA), 2,2-дилинолеил-4-диметиламинометил-[1,3]-диоксолан (DLin-K-DMA), производные фосфатидилсерина [1,2-диолеил-sn-глицеро-3-фосфо-L-серин, натриевая соль (DOPS)], холестерин}, белки (например, альбумин, желатины, ателлоколлаген) и пептиды (например, протамин, PepFects, NickFects, полиаргинин, полилизин, CADY, MPG).
В контексте изобретения конъюгатные группы выбирают из группы, состоящей из нацеливающих частей, повышающих стабильность частей, усиливающих захват частей, усиливающих растворимость частей, улучшающих фармакокинетику частей, улучшающих фармакодинамику частей, повышающих активность частей, репортерных молекул и лекарственных средств, где эти части могут представлять собой пептиды, белки, углеводы, полимеры, производные этиленгликоля, витамины, липиды, полифторалкильные части, стероиды, холестерин, флуоресцентные части и радиоактивно меченые части. Конъюгатные группы необязательно могут быть защищенными и они могут быть связаны с олигонуклеотидом по изобретению прямо или через двухвалентный или поливалентных линкер.
Конъюгатные группы включают части, которые усиливают нацеливание, захват, растворимость, активность, фармакодинамику, фармакокинетику или которые снижают токсичность. Примеры таких групп включают пептиды (например, глутатион, полиаргинин, пептиды RXR (см., например, Antimicrob. Agents Chemother. 2009, 53, 525), полиорнитин, TAT, TP10, pAntp, полилизин, NLS, пенетратин, MSP, ASSLNIA, MPG, CADY, Pep-1, Pip, SAP, SAP(E), транспортан, буфорин II, полимиксин B, гистатин, CPP5, NickFects, PepFects), vivo-porter, белки (например, антитела, авидин, Ig, трансферрин, альбумин), углеводы (например, глюкоза, галактоза, манноза, мальтоза, мальтотриоза, рибоза, трегалоза, глюкозамин, N-ацетилглюкозамин, лактоза, сахароза, фукоза, арабиноза, талоза, сиаловая кислота, гиалуроновая кислота, нейрамидиновая кислота, рамноза, квиновоза, галактозамин, N-ацетилгалактозамин, ксилоза, ликсоза, фруктоза, манноза-6-фосфат, 2-дезоксирибоза, глюкал, целлулобиоза, хитобиоза, хитотриоза), полимеры (например, полиэтиленгликоль, полиэтиленимин, полимолочная кислота, поли(амидоамин)), производные этиленгликоля (например, триэтиленгликоль, тетраэтиленгликоль), водорастворимые витамины (например, витамин B, B1, B2, B3, B5, B6, B7, B9, B12, C), жирорастворимые витамины (например, витамин A, D, D2, D3, E, K1, K2, K3), липиды (например, пальмитил, миристил, олеил, стеарил, батил, глицерофосфолипид, глицеролипид, сфинголипид, церамид, цереброзид, сфингозин, стерин, пренол, эруцил, арахидонил, линолеил, линоленил, арахидил, бутирил, сапеинил, элаидил, лаурил, бегенил, нонил, децил, ундецил, октил, гептил, гексил, пентил, DOPE, DOTAP, терпенил, дитерпеноид, тритерпеноид), полифторалкильные части (например, перфтор[1H,1H,2H,2H]-алкил), одобренные лекарственные средства с ММ<1500 Да, которые обладают аффинностью к конкретным белкам (например, NSAID, такие как ибупрофен (больше описано в патенте США 6656730, включенном в настоящее описание в качестве ссылки), антидепрессанты, противовирусные средства, антибиотики, алкилирующие средства, амебицидные средства, аналгетики, андрогены, ингибиторы ACE, снижающие аппатит средства, антациды, противогельминтные средства, антиангиогенные средства, антиадренергетики, антиангинальные средства, антихолинергетики, антикоагулянты, противосудорожные препараты, противодиабетические средства, противодиарейные средства, антидиуретики, антидоты, противогрибковые средства, противорвотные средства, средства против головокружения, противоподагрические средства, антигонадотропные средства, антигистамины, антигиперлипидемические средства, антигипертензивные средства, противомалярийные средства, средства против мигрени, средства против злокачественной опухоли, антипсихотические средства, противоревматические средства, антитиреоидные средства, антитоксины, противокашлевые средства, анксиолитические средства, контрацептивы, стимуляторы ЦНС, хелаторы, сердечно-сосудистые средства, противозастойные средства, дерматологические средства, диуретики, отхаркивающие средства, диагностические средства, желудочно-кишечные средства, анестетики, глюкокортикоиды, антиаритмические средства, иммуностимулирующие средства, иммунодепрессивные средства, слабительные средства, лепростатические средства, метаболические средства, средства, действующие на органы дыхательной системы, муколитики, мышечные релаксанты, нутрицевтические средства, сосудорасширяющие средства, тромболитические средства, утеротонические средства, сосудосуживающие средства), природные соединения с ММ<2000 Да (например, антибиотики, эйкозаноиды, алкалоиды, флавоноиды, терпеноиды, кофакторы ферментов, поликетиды), стероиды (например, преднизон, преднизолон, дексаметазон, ланостерин, холевая кислота, эстран, андростан, прегнан, холан, холестан, эргостерин, холестерин, кортизол, кортизон, дефлазакорт), пентациклические тритерпеноиды (например, 18β-глицирретиновая кислота, урсоловая кислота, амирин, карбеноксолон, эноксолон, ацетоксолон, бетулиновая кислота, азиатиковая кислота, эритродиол, олеанолевая кислота), полиамины (например, спермин, спермидин, путресцин, кадаверин), флуоресцентные части (например, FAM, карбоксифлуоресцеин, FITC, TAMRA, JOE, HEX, TET, родамин, Cy3, Cy3,5, Cy5, Cy5,5, CW800, BODIPY, AlexaFluors, Dabcyl, DNP), репортерные молекулы (например, акридины, биотины, дигоксигенин, (радио)изотопно меченые части (например, с 2H, 13C, 14C, 15N, 18O, 18F, 32P, 35S, 57Co, 99mTc, 123I, 125I, 131I, 153Gd)) и их комбинации. Такие конъюгатные группы могут быть присоединены прямо к соединениям по изобретению или через линкер. Этот линкер может быть двухвалентным (обеспечивающим конъюгат 1:1) или поливалентным, обеспечивающим олигомер с более чем одной конъюгатной группой. Методики связывания такой конъюгатной группы, либо прямо, либо через линкер, с олигомером по изобретению известны в данной области. Также в контексте изобретения находится применение наночастиц, с которыми ковалентно связаны олигонуклеотиды по изобретению, таким образом, такие конструкции называют сферическими нуклеиновыми кислотами (SNA), например, как описано в J. Am. Chem. Soc. 2008, 130, 12192 (Hurst et al., включенная в настоящее описание в качестве ссылки).
В настоящем документе и в его формуле изобретения глагол "содержать" и его спряжения используют в неограничивающем значении для обозначения того, что объекты после этого слова включены, однако объекты, конкретно не упомянутые, не исключены. Кроме того, глагол "состоять из" может быть заменен на "по существу состоять из", что означает, что олигонуклеотид или композиция, как определено в настоящем описании, могут содержать дополнительный компонент(ы) помимо компонентов, конкретно указанных, причем указанный дополнительный компонент(ы) не изменяет уникальной характеристики изобретения. Кроме того, указание на элемент в единственном числе не исключает возможности того, что присутствует более одного элемента, если контекст явно не требует, чтобы существовал один и только один из элементов. Таким образом, форма единственного числа обычно означает "по меньшей мере один".
Слова "приблизительно" или "примерно", когда их используют применительно к числовой величине (приблизительно 10), предпочтительно означают, что величина может представлять собой данную величину, равную 10, или отличается менее чем на 1% от этой величины.
Каждый вариант осуществления, как указано в настоящем описании, может быть комбинирован с другим, если нет иных указаний. Все патентные и литературные ссылки в настоящем описании включены в настоящее описание в качестве ссылки в полном объеме.
Следующие примеры представлены только для иллюстративных целей, и они не предназначены для ограничения объема настоящего изобретения никоим образом.
Примеры
Таблицы 1-3
Перечень возможных комбинаций экзонов в транскрипте гена DMD, для которых экзон U (вышележащий) имеет непрерывную открытую рамку считывания с экзоном D (нижележащий), если экзоны с U+1 (первый экзон) по D-1 (второй экзон), и любые экзоны между ними, удалены из транскрипта.
29,30,31,32,33,34,35,36,37,38,39,40,41,
42,43,45,47,49,50,52,54,56,58,60,61,68,70
30,31,32,33,34,35,36,37,38,39,40,41,42,43,45,47,49,
50,52,54,56,58,60,61,68,70
30,31,32,33,34,35,36,37,38,39,40,41,42,
43,45,47,49,50,52,54,56,58,60,61,68,70
43,45,47,49,50,52,54,56,58,60,61,68,70
32,33,34,35,36,37,38,39,40,41,42,43,45,
47,49,50,52,54,56,58,60,61,68,70
33,34,35,36,37,38,39,40,41,42,43,45,47,
49,50,52,54,56,58,60,61,68,70
33,34,35,36,37,38,39,40,41,42,43,45,47,
49,50,52,54,56,58,60,61,68,70
35,36,37,38,39,40,41,42,43,45,47,49,50,
52,54,56,58,60,61,68,70
54,56,58,60,61,68,70
37,38,39,40,41,42,43,45,47,49,50,52,54,
56,58,60,61,68,70
38,39,40,41,42,43,45,47,49,50,52,54,56,
58,60,61,68,70
38,39,40,41,42,43,45,47,49,50,52,54,56,
58,60,61,68,70
39,40,41,42,43,45,47,49,50,52,54,56,58,
60,61,68,70
40,41,42,43,45,47,49,50,52,54,56,58,60,
61,68,70
42,43,45,47,49,50,52,54,56,58,60,61,68,
70
43,45,47,49,50,52,54,56,58,60,61,68,70
45,47,49,50,52,54,56,58,60,61,68,70
47,49,50,52,54,56,58,60,61,68,70
49,50,52,54,56,58,60,61,68,70
50,52,54,56,58,60,61,68,70
52,54,56,58,60,61,68,70
54,56,58,60,61,68,70
56,58,60,61,68,70
58,60,61,68,70
60,61,68,70
61,68,70
68,70
Перечень областей экзонов: участки последовательности с (частично) высокой идентичностью или сходством последовательности (по меньшей мере 50%) между двумя различными экзонами дистрофина, идентифицированные в качестве наилучшего попарного выравнивания с помощью EMBOSS Matcher с использованием параметров по умолчанию (матрица: EDNAFULL, штраф за пропуск: 16, штраф за продолжение пропуска: 4) (http://www.ebi.ac.uk/tools/psa/emboss_matcher/nucleotide.html). Пропускание этих экзонов, и предпочтительно любых экзонов между ними, может привести к транскрипту DMD в рамке считывания (как в таблице 1)
19
||.|.||||||.||
AGCUGAGAAGUUCA
18
21
||..|||....|||| |||||..|..|.||||.||
ACAAAUCAUUUUAAGCAAGUCUUUUCUGAUGUGCA
20
50
|||.|.||||.|..|||| ||..|.||||
UUUACUUCAAGAGCUGAGGGCAAAGCAGCC
22
52
|.|.|.||.|||||..|||||
GCAACAAUGCAGGAUUUGGAA
24
58
||||.|.|.||||||
CAAGAGGGAAUUGAA
26
10
|.||||||.|||.|
CAAGCACAAGGAGA
28
12
|.|.|.|||.||.|...|.|.||.|||||
GACUGGCUAACAAAAACAGAAGAAAGAAC
30
13
|||||.|||.|.|.|.|..||
UCUAGAACAAGAACAAGUCAG
32
14
|.|||..||.||.|..|..|.||.|||.||
AGACAUCCUUCUCAAAUGGCAACGUCUUAC
34
15
|.|||..||.||.|.|.|||.||
UUUAGUGCAUGGCUUUCAGAAAA
36
16
|||.|...||.|||.|.| |||.|.|||.|||
UAUUCACUCAAACAAGAU-CUUCUUUCAACAC
38
18
|||||.|..|||||||
GAUAUAACUGAACUUC
40
20
||||.|.||.||.||.|||
CACCCCAUCAGAGCCAACA
42
22
|||| |||||||..||.|||
CUUAGUGUCACCGACUAUGA
44
23
|||.|||.||||.|||
UAUCUCAGCACCACUG
46
25
|||.|||||. |||.|| |||..|||
CAGUGAUAUUCAGACAA-UUCAGCCC
48
26
||||||||.|.. ||||.||||
UAAGCCUCCAGA-AAGAUCUAU
50
27
||.|||..||.|.||.|||
ACUACCAGUGGCUCUGCAC
52
28
|||..||||..||..|||
GGAGCUGAGGAAAUCUCU
54
29
|||.|..|||||.|..|..|||.|.|
AGGAACUUGAGACAUUUAAUUCUCGU
56
30
|||||| ||.||||....|||.|.|.|.| |.|.|.|||.||..||.||
CAGUCU-GCCCAGGAGACUGAAAAAUCCU-UACACUUAAUCCAGGAGUC
58
31
|.|||||.|||.| ||.||||...|||.|..||
GAUCAGUUUAGAA----GAAAUGAAGAAACAUAAUC
60
32
||.||||...||.|..|.|.|...||||...|.|||||
UUAUUCCAGAAACCAGCCAAUUUUGAGCAGCGUCUACA
62
33
|||.|||. ||.|||..|||||
CCCAAAGAACUUGAUGAAAGAG
64
34
|.||.|||...|||.||.| |||.||||...|||....|.|...|||.||..|||.| |||||
GAGAAAUGCUUGAAAUUGU-CCCGUAAGAUGCGAAAGGAAAUGAAUGUCUUGACAGAAUGGCUG
66
35
||..||.|||||.|.||.|||||..|||
AGAGUAUCACAGAGGUAGGAGAGGCCUU
68
36
||||.||..||||. |||| ||.||||||
UCAGGCUGACACACUUUUGGAUGAAUCAGAGAA
70
38
||||.||.|.|| |||||...|.||.|.|||
CAGUUUAACUCA--GAUAUACAAAAAUUGCUU
72
40
|||||||.|.||.||
CAGGCUGAUGAUCUC
74
41
|||.|||.||.||
CCAACUCAGAUCC
76
42
||..||..|.|||||||
GAAGUGGAACAACUUCU
78
44
|.|||.|||.|||.||
GCGAUUUGACAGAUCU
80
46
|||||..|..||.||.|||.|.|..|||.||.||
GUCAGAAUUUCAAAGAGAUUUAAAUGAAUUUGUU
82
47
||||.||||||..||
UCCCAUAAGCCCAGA
84
48
|.|||.||||||.|.||| |.|.|||.|
GUUUCCAGAGCUUUACCUGAGAAACAAG
86
49
|.|||..|||. ||...||||.||..|||..||.|
GUUCAAGCUAAACAACCGGAUGUGGAAGAGAUUUU
88
51
|.|.|.|||..|...||.|||.|..||.|||||||
CUCUGGCAGAUUUCAACCGGGCUUGGACAGAACUU
90
53
|.|||||..|||||
CAACACAAUGGCUG
92
57
|||| ||||.....|.||...||||||||
GUCU-GCACCUUUCUCUGCAGGAACUUCU
94
59
|||||||...|.||||..||
CAGGCUGAGGAGGUCAAUAC
96
60
||.||||.|||.||.|
CAGCUCUCACCGUAUA
98
12
||||.|.||||...|.|||..|||..|.||....|||.||
UGAUCUUGAAGACCUAAAACGCCAAGUACAACAACAUAAG
100
13
||||..||.|.|||||.|||.|
UCAAGAAGAUCUAGAACAAGAA
102
14
||||||.||.||||.|.|.. |..|.|| |||.||||||
CAAACAUCUGUAGAUGGACAGAAGACCGCUGGGUUCUUU
104
15
|.||.|..|.|.|||.|.|||||..|.||||
AAAGAUCAAAAUGAAAUGUUAUCAAGUCUUC
106
16
|.|.||.|..||.|.|..|..|||| ||..||||.||||
ACUCAAACAAGAUCUUCUUUCAACA-CUGAAGAAUAAGU
108
18
|||||.||.||. |.||||..|.|..|.||..|||
AUUUGCAAUCUUUCGGAAGGAAGGCAACUUCUCAG
110
20
|||||||| ||.|.||||.||.|
AUCAAACAAGCCUCAGAACAACU
112
22
|||||.||||.|.||
GACACUUUGCCACCA
114
23
||||.|.|.||.|.| .||||||.|||..||.||||||.|
UGAAAUUAGCCGGAA---AUAUCAAUCAGAAUUUGAAGAAAU
116
25
|.||.|| |||.|...|||||.|.|||.|.|...|||...|||
AGACUUGAGACAGAACUCAAAGAACUUAACACUCAGUGGGAUC
118
26
||.|.||.||...|..|.|||||...|.||||||
CUCCAGAUGAAUUACAGAAAGCAGUUGAAGAGAU
120
27
|.||||..||||||.|
AGAGCUAAAGAAGAGG
122
28
|.||.|||| |..|.||||||..|. ||.|.||||||..||.....|||....|..|||| |.|||||.|..||
AUACUUGGA--GAAAGCAAACAAGUGGCUAAAUGAAGUAGAAUUUAAACUUAAAACCACUGA--AAACAUUCCUGGC
124
29
|||||.| ||.||||.|.|.||.|
CUAAUCAAUGAGGAACUUGAGACA
126
30
|||||| ||.|||||||.|.|| |.||.||..||..|||||.....|||||...||...||||
GACAAG-CAGUUGGCAGCUUAU--------AUUGCAGACAAGGUGGACGCAGCUCAAAUGCCUCAGGAAG
128
31
||||||||||. ||.|||.|...|.|||. |||||.||
GACAAGUCAUG----AGAUCAGUUUAGAAGAAAUGAAGAAA
130
32
|||||..|.|..|..|.|..|.||||.||.|. ||.|.|.. ||||.|..|..|.||||.|| |.|.||| ||||||.||..||
ACAAGAAAGUAAGAUGAUUUUAGAUGAAGUGAAGAUGCACUUGCCUGCAUUGGAAACAAAGAG-UGUGGAACAGGAAGUAGUACAGU
132
33
|||.||||||.|||||| |||||
UGAAGUGGAAAUGGUGAUAAAGAC
134
34
|||..|||.|||||.||...|| |||.|||
CUACAGAUAUGGAAUUGACAAAGAGAUCAG
136
35
||||||.|||.| ||||||.|
AAACAGUUUUGGGCAAGAAGGA
138
36
||.||||.|||.|..|||.....|| ||||||
AAACACAUGGAAACUUUUGACCAGAAUGUGGA
140
37
|||.|||..|||||
GAAUUAAGACUGGA
142
38
|||||.|| .||||.|..|.|.|..|||.|.||.||.||.|
UGAAGGAA--UUGGAGCAGUUUAACUCAGAUAUACAAAAAUU
144
39
||||.||.|.||.|.||..|||.|
AUUGUUGCAAAGAGGAGACAACUU
146
40
|||||.|.||..|.|...|.|||| |||.||||||.||.| ||||.|...||..||||
UGAGGUCUCAAAGAAGAAAAAAGGCUCUAGAAAUUUCUCAUCA-GUGGUAUCAGUACAAGA
148
41
|||||...||....|.|.|||....|||.|.|| |||.|.||||.||
GCUGAAUGCAGUGCGUAGGCAAGCUGAGGGCUUGUCUGAGGAUGGGG
150
42
|||||||||.| |.|||||.|.||..|| |||.....|..|.|.| .|.|||.|..||||..||..|.| |.|||...|||||.| |||| |||||...||||...|||...| |||
CUGAAGACAUG-CCUUUGGAAAUUUCUU-AUGUGCCUUCUACUUA--UUUGACUGAAAUCACUCAUGUCUCACAAGCCCUAUUAGAAGUGGAACAACUUCUCAAUGCUCCUGACCUCUGUGCU//
|||..|...|| ||||.|||
//AAGGACUUUGA-AGAUCUCU
152
44
||||.|||....|.|.|| ||||..|.|.||| ||||||.......||||| |||..||...|| |.|...|.|.|||.||.|..||..||||...|..|||.||.|..
CGUUUUCAUUAUGAUAUA-AAGAUAUUUAAUCAGUGGCUAACAGAAGCUGA--ACAGUUUCUCAG-AAAGACACAAAUUCCUGAGAAUUGGGAACAUGCUAAAUACAAAU
154
46
|||.|.|.||.|.|.|. |.|||||.||.||||.|.||| ||.||||.||.|
AAGAAUAUCUUGUCAGAAUUUCAAAGAGAUUUAAAUGAAUUUGUUUUAUGGUUG
156
47
||||||..|.||||||.||
AAGCUCAAGCAGACAAAUC
158
48
|.|||||||. ||.|.|||.|.||.|||||.|
UUGAAGCUCA--AAUAAAAGACCUUGGGCAGCU
160
49
||.|.||||.||....|..|...|.|.|||.|.|.|.|.|||||..|.||.||
GAAACUGAAAUAGCAGUUCAAGCUAAACAACCGGAUGUGGAAGAGAUUUUGUC
162
51
|||.||||||..|.|.|.|......|||| |.|.|.||||.|.|..||.|| ||||.| ||.|| |||||| |.|||||| ||..|.....|..||.||||..|.|.|||
UUCCUUGAUGUUGGAGGUACCUGCUCUGG-CAGAUUUCAACCGGGCUUGGACAGAACU--UACCGACUGGCUU-UCUCUGCUUGAUCAAGUUAUAAAAUCACAGAGGGUGAU
164
53
||.|||.|||. |||.|...|||.|..||..||||.||..||||.|
GAAAGAAUUCAGAAUCAGUGGGAUGAAGUACAAGAACACCUUCAGA
166
55
|.|||||.||||||
CUGCUUUGGAAGAA
168
57
|||||||||||...|...|||.|||
CAUUUGGAAGCCAGUUCUGACCAGU
170
59
||..||.|||||||.|.|
CUGCCUCCUGAGGAGAGA
172
60
||..||||||||.|||.||
CAGAUGGAAGCUUCUGCAG
174
12
|||||||..|||| ||| ||||....||||.|.||||.|||....|.|.....|.|..||.|..|||||..|| |.||....|.|||.|.|.||
AUCAGAAACUGAAAGAGUUGAAUGACUGGCUAACAAAAACAGAAGAAAGAACAAGGAAAAUGGAGGAAGAGCCUCUUGG-ACCUGAUCUUGAAGACCUAA
176
13
|||||||..|||| .|||.|||||....|.|.|||...||.|.|. ||.|.|||
AGAAGAUCUAGAA---CAAGAACAAGUCAGGGUCAAUUCUCUCACUCACAUGGUGG
178
14
||||...||||....|||.|.|..||||.||
GAUGGGCAAACAUCUGUAGAUGGACAGAAGA
180
15
|.||||||.|.|||||..|.|. |.||||||. ||.|..|.|..|.| |.|||||...||||. .|...||.|.||.|.|.||||||
UUUCAGAAAAAGAAGAUGCAGU-GAACAAGAUUCACACAACUGGCUU----UAAAGAUCAAAAUGA--AAUGUUAUCAAGUCUUCAAAAAC
182
16
||||||..|.|.|.||.|.|||.| ||.||...||.||. ||||||
AAGAAAAAGCAAUCCAUGGGCAAACUGUAUUCACUCAAA-CAAGAU
184
18
|||.||.||.|||||.....||||||
AGGCAACUUCUCAGACUUAAAAGAAA
186
20
|.|||.|.|...|||||.|.| |||..||||.||....|| |||.||... |.||||||||.|
CCGGUGGAUCGAAUUCUGCCAGUUGCUAAGUGAGAGACUU--AACUGGCUGGAGUAUCAGAACA
188
22
||||||||.|..||.|
CCAUCAGGACAUGGGU
190
23
||||....|.|.|||||..| |||||||..||||||||.|||.|.||....|.|||.|. ||||||
CUGAAAUUAGCCGGAAAUAUCAAUCAGAAUUUGAAGAAAUUGAGGGACGCUGGAAGAAGC-UCUCCU
192
24
|||..|||||.|. ||.||.|||||.||||.|
GAUUCAGAAAUUC---UAAAAAAGCAGCUGAAAC
194
25
||..||.||||||. ||||||..|. |||......|.||..| |.|||..|...||||||||.||||| |||||.|.|
GUCAGUGAUAUUCAGACAAUUCAGCCCAGUCUAAACAGUGUCA-AUGAAGGUGGGCAGAAGAUAAAGAA--UGAAGCAGA
196
26
|||.||.|| ||||||| ||||.|..|| ||.|..|||..|||.||.|||
CAGAAAGAUCUAUCAGA-GAUGCACGAA-UGGAUGACACAAGCUGAAGAA
198
27
||||.||.|......||..|.|||.|.|..||||.|||||.|.|
AAGAAGAGGCCCAACAAAAAGAAGCGAAAGUGAAACUCCUUACU
200
28
|.||.|||.|||| ||||.||.|.|||
GGAGCUGAGGAAAUCUCUGAGGUGCUAGA
202
29
||..|||||.|||| |...||.||||.|..|.||..|..||.|..|.| |.|| ||.|||..|...|.|||.||
GCAUAUUGGCACAG--ACCCUAACAGAUGGCGGAGUCAUGGAUGAGCUA-AUCAAUGAGGAACUUGAGACAUUUAA
204
31
||.||||||.||..||..|....|||||...| ||||||| |||||.| ||..|.|||..|||
AAUCUGAUUUGACAAGUCAUGAGAUCAGUUUA-GAAGAAA-UGAAGAAACAUAAUCAGGGGAA
206
32
|||||.||..|||. |..|||||||||||
AUUGGAAACAAAGA-GUGUGGAACAGGAA
208
33
||||.|.|.|| ||| ||||| .|||||||..||..|..|||.||
AAAAGUCUGAGUGAAG-UGAAG--UCUGAAGUGGAAAUGGUGAUAAA
210
34
|||||..|..|||.. |||||.||.|||||.||
UGGAAUUGACAAAGAGAUCAGCAGUUGAAGGAA
212
35
||||..||||||.||||
AGAUUGAGAAACAGAAG
214
36
|||||| ||..|||||||...||.| ||.||.|||.|
AUCAGA-GAAAAAGAAACCCCAGCA-AAAAGAAGACG
216
37
||.|||||| |||||
AUCGAUUUG-CAGCC
218
38
||||||....|||......||..|||||.|.||..||..|.|||||||
AAAAUUGCUUGAACCACUGGAGGCUGAAAUUCAGCAGGGGGUGAAUCU
220
39
||||...|| ||.||.|.||| ||.||.|||||.||
AGAAAGCGAGAGGAAAUAAAGAUAAAACAGCAGCUG
222
40
|||||||||| |||. ||| |||||||.||.|
AGUACAAGAGGCAGGCUGAUGAUCUCCUGAAAU
224
41
||||.|.|||.|.|..||.|||| ||||.| ||||.||.|||||
GAAAUUGAUCGGGAAUUGCAGAA---GAAGAA--AGAGGAGCUGAAU
226
42
|||.|||||..|||.||
UAUUAGAAGUGGAACAA
228
44
|||..|||.||..|.|.||..|.| ||||....|||.|||.||..|.|..|.||||..||| |||||
ACAGAAGCUGAACAGUUUCUCAGA---AAGACACAAAUUCCUGAGAAUUGGGAACAUGCUAAAUACAAA
230
46
||||.||||.||.|.||.. |.||..||.|||||
GAAGAACAAAAGAAUAUCU-UGUCAGAAUUUCAA
232
47
|||||||..|||| ||||..|..|.||| ||||||..|||||||
CAGAAGAGCAAGAUAAACUUGAAAAUAAGCUCAAGCAGACAAAUCUCC
234
48
|.||||...||||| ||.|..||||.||| ||||..||||
ACCUGAGAAACAAGGAGAAAUUGAAGCUC--AAAUAAAAGA
236
49
|.|||.|.||.|..|....|||.|.||.||.|.||.||..|||||
GAAACUGAAAUAGCAGUUCAAGCUAAACAACCGGAUGUGGAAGAG
238
51
|.|||.||.|.|..|.|||..|.|.||||||
AGGAAACUGCCAUCUCCAAACUAGAAAUGCC
240
53
||||....||||||...|..|.| ||||||||||||.||
GAAAGAAUUCAGAAUCAGUGGGA--UGAAGUACAAGAACA
242
55
|||.||....|..|.||||.||||||.||||||.|
AGAAGACUCCAAGGGAGUAAAAGAGCUGAUGAAAC
244
57
||.||..|.|| |||||| ||..|.||..|||.||.| ||||.| |..||.|||||
GGCAGGCACCU-AUUGGA---GGCGACUUUCCAGCAGUUCAGAAGCAGAACGAUGUACA
246
59
|||...|.|.||.||.||.|||..||.. |.|.|..|||||.|||.|.||.|||||. ||.|||...|..|.|.||| |||.|.||||||...|.|.||...||..|.
UCAAUACUGAGUGGGAAAAAUUGAACCU----GCACUCCGCUGACUGGCAGAGAAAAAUAGAUGAGACCCUUGAAAGACUCCAGGAACUUCAAGAGGCCACGGAUGAGCUGG ACC//
|||||.||.|.....||.|||..|.|.||
//UCAAGCUGCGCCAAGCUGAGGUGAUCAAG
248
60
|||.||..|.||||||.|
CCUGAACACCAGAUGGAA
250
17
|||.|.|..|.|||||||
ACAGACAACUGUAAUGGA
252
43
|.||||||||.. |.|||.|...||||||
GUUAACAAAAUGUACAAGGACCGACAAGG
254
45
||||||||||.|..||.| ||.|.|||||.| |||||
CAAAAACAGAUGCCAGUAUUCUACAGGAAAAAUUG-GGAAG
256
54
|.|||.|||.|.|.||||..|||.. ||||.||
UAACAGAGAAUAUCAAUGCCUCUUGGAGAAGCA
258
56
||.||||| |||...|||.||.|.|.||.||. ||.||...||. |..|||||||...|||.|.|
GACCUCCAAGGUGAAAUUGAAGCUCACACAGAUGUUUAUCACAACCUGGAUGAAAACAGCCAAAAAAUC
260
15
||||.|||||| |||||. ||||.|||
UCACACAACUG--GCUUUA-AAGAUCAA
262
16
|||..||||||||| |||||.|||
AAGCGGAUCUAGAA-AAGAAAAAG
264
18
|||||...|||....|.||||||.|| |..||..|||||.||.|.||
AAUCUUUCGGAAGGAAGGCAACUUCUCAGACUUAAAAGAAAAAGUCAA
266
20
|.|.|.|||||...|.| |||||.|||||
AACAAUUGGAGCAGAUGACAACUACUGCU
268
22
||||||...|.|..|||||
AGUCAGAAACCAAACUCUC
270
23
||||.|..|||.|||... |||||.|||...|| ||.|.||.||.||.|
GCUUUACAAAGUUCUCUG-CAAGAGCAACAAAGUGGCCUAUACUAUCUC
272
24
|.|||| |||..|.||||||||
AAAUGGAUGGCUGAAGUUGAUG
274
25
|||||||..||||..|.....||.|||.||
AGAAGAUAAAGAAUGAAGCAGAGCCAGAGU
276
26
||.||..||||||||
CUCCAGAAAGAUCUA
278
27
|.|||||||.....|||||.||.|||..|..||.||....||.|||.|
UAAAGAAGAGGCCCAACAAAAAGAAGCGAAAGUGAAACUCCUUACUGA
280
28
||||.|||....||||..|| |..||.|..||.|.||..|||.|| |||... ||||.|.||.||||
CAUGUUGGCAUGAGUUAUUGUCAUACUUGGAGAAAGCAAACAAGUG--GCUAAAUGAAGUAGAAUUUAA
282
29
|||||..|.|.||.|..||||
CAUGGAUGAGCUAAUCAAUGA
284
30
|||.|.|||.|..|.|||.||.|.|| |.|..||...||.|. |||||.||
UCAUUGACAAGCAGUUGGCAGCUUAU-AUUGCAGACAAGGUGGACGCAGCU
286
31
||||||..| |||.||..|.|| ||||||
AGAAGAAAU-GAAGAAACAUAA-UCAGGG
288
32
|||||.|.|||..|| ||..|.|||..||| |||.||||||
GAUGAUUUUAGAUGAAGUGAAGAUGCACUUGCCUGCAUUGGAA
290
34
|||.||.|.|.|.|||||
GCAGCUACAGAUAUGGAA
292
35
||||.||||..|.||..||.|..|.||.....|.||...|.||||..|..|.|..|.||.. ||||..|...|..|||..|..|.|.|...|.||. |||||||..||||.|
UCAAAAAGAGAUUGAGAAACAGAAGGUGCACCUGAAGAGUAUCACAGAGGUAGGAGAGGCC-UUGAAAACAGUUUUGGGCAAGAAGGAGACGUUGG--UGGAAGAUAAACUCA
294
36
|.||||||||. ||||..|.|.|||..|.||.... |||||....||||||
UGGAUGAAUCA----GAGAAAAAGAAACCCCAGCAAAAAGAAGACGUGCUUAAG
296
37
||.||||...|.||.||.|||.|
GACUCUACACGUGACCAAGCAGC
298
38
||||..||.|.|||.|..|||
AUCUGAAAGAGGAAGACUUCA
300
39
||..||.|.||.|||.||.|.|. |||.||.|||.|...||| |.|||.|||
GAGAAAGCGAGAGGAAAUAAAGAUAAAACAGCAGCUGUUACAGACAAAACAUAA
302
40
||.|||..||.|||||.|| |||.|| ||.|.||||| ||| |||||...|||..|.||..|..|..|| ||||.|.. ||.||||
AGGUCUCAAAGAAGAAAAA---AGGCUCUAGAAAUUUCUCA-UCA----GUGGUAUCAGUACAAGAGGCAGGCUGAUGAUCUCCUGAAAUGCU
304
41
|||||||...||.||..|.|.|||..|.|||
AGUGGAGCCAACUCAGAUCCAGCUCAGCAAG
306
42
|||.|||.|.|. |||.|||.||..||....||| |||||||
UGAAGAAACGAUGAUGGUGAUGACUGAAGACAUGCCUUUGGAA
308
44
||.|..|.|||| |||||..|.|| |..|||.|.|.||..|.|||| ||||.||
AGAUAUUUAAUC-AGUGGCUAACAGAAGCUGAACAGUUUCUCAGAAAGACACAAAUU
310
46
||..|||||.|.||.||.||||
CUAAAAGAAAAGCUUGAGCAAG
312
47
|||||.|.|.|||..|. |||.|||||..||.|
GUGCUCCCAUAAGCCCAGAAGAGCAAGAUAAACU
314
48
||.|.||.|.|..||.||||||||.|||
AGAGCAGUUAAAUCAUCUGCUGCUGUGG
316
49
|.|.|||||.|.||..|||
UUGUACAAGGAAAAACCAG
318
51
|.||||.|.....|.|||||..| ||||.|.||.|.|||. ||.|.|||.||||...|..||.|..|...|.||||
GAUCAAGUUAUAAAAUCACAGAG-GGUGAUGGUGGGUGAC-CUUGAGGAUAUCAACGAGAUGAUCAUCAAGCAGAA
320
53
|||..||.|||||||||
GAUGAAGUACAAGAACA
322
55
|||||||||||||||
GCUGCUUUGGAAGAA
323
57
|||.||.||.|| ||||..|||||.|
UUCCAGCAGUUC-AGAAGCAGAACGA
325
59
||.||||.|...|.||...||.|..||||.||.||..||
GGCGAUCUCCUCAUUGACUCUCUCCAAGAUCACCUCGAG
327
60
|.||.|||..||.||.||||. |||.|.||
GAAAUUGCGCCUCUGAAAGAG-AACGUGAG
329
15
|.|.|||.|.|||.||.|
AUGGCUUUCAGAAAAAGA
331
16
|||.||||||||||
AUCCAUGGGCAAAC
333
18
|.||.||.|.|||| ||||.|.||||.|
UCCUGAAUUUGCAA--UCUUUCGGAAGGA
335
20
||.|||.|||| |..|.|||||.|
CAGAUGACAAC-UACUGCUGAAAA
337
22
|||..|||||.| |||.|... ||||.|.|.||||
UAUCAGGAGACC-AUGAGUGC-CAUCAGGACAUGG
339
23
||||||| |..|| |||..||..|||||
UUUACAA-AGUUC--UCUGCAAGAGCAAC
341
24
||||.||...|..|..|.|...|.||.||.||||||.|
AAGAAAUGGAUGGCUGAAGUUGAUGUUUUUCUGAAGGA
343
25
||.||.|||.|||||||
UGAAGGUGGGCAGAAGA
345
27
|||..|.|||..||||||||
AAAGUGAAACUCCUUACUGA
347
28
|||||||..|| ||.|||.|
GCAAACAAGUGGCUAAAUGAA
349
29
||.||||.||.||
AUGGAUGAGCUAA
351
30
|||.||.||......|||.||.|||||
CAGGAGUCCCUCACAUUCAUUGACAAG
353
31
|||.|||||.|||
UUUGACAAGUCAU
355
32
|.||||||.|..|||||
UUUAGAUGAAGUGAAGA
357
33
||.||||.|.|||||
AAAGUCUGAGUGAAG
359
34
||||| ||.||||| ||.|||
AAAUG--AAUGUCUUGACAGAA
361
35
|||||||..|.|....||||||
CAGAAGAGUGGUUAAAUCUUUU
363
36
||.||||.|||.|.|.||
CACAUGGAAACUUUUGAC
365
37
||||||..|||..|||.|. |.|..||...|||||.|
AUCGAUUUGCAGCCAUUUC-ACACAGAAUUAAGACUG
367
38
|||.|||..|.||....|.|.|.||||
UCUGAAAGAGGAAGACUUCAAUAAAGA
369
39
||||||.....||.|..|.||.|||| ||..||...||..||..||..|||||
GAAGACAAUGAGGGUACUGUAAAAGA-AUUGUUGCAAAGAGGAGACAACUUAC
371
40
|.||||.|..||||||.|
AGGUCUCAAAGAAGAAAA
373
41
||||.||.|| |.|||||
GGGAAAUUGA-GAGCAAA
375
42
||||||| ||||...||||
AAGACAUGCCUUUGGAAAU
377
44
|||||||..|||.||..|.|.||
AAAUGGCGGCGUUUUCAUUAUGA
379
46
||||.|.|||.||||
UAGAAGAACAAAAGA
381
47
||||||||.....|..|...||||.||.||
UUCUCAAACAAUUAAAUGAAACUGGAGGAC
383
48
|.|.|||.||..||...|||.||.....|||.||||
AGUUGGAAAUUUAUAACCAACCAAACCAAGAAGGAC
385
49
|.|||||. |||.||||.|.|||.|.|
AAGGGCAG-CAUUUGUACAAGGAAAAA
387
51
|||||||||. |||| ||||.||..|.|.|..||.....||.|.||||..||
UGGACAGAACUUACCGACUGGCUUUCUCUGCUUGAUCAAGUUAUAAAAUCACA
389
53
||||.|....|||||||...|||||..||..|| ||||...|.|.||||
UGGGAUGAAGUACAAGAACACCUUCAGAACCGGAGGCAACAGUUGAAUGAA
391
55
|||.||.|||.|||.|||.|.|
CAACUGCCAAUGUCCUACAGGA
393
57
|.|||..|||.||....|..|.|.|||.|.|||||
CAUUUGGAAGCCAGUUCUGACCAGUGGAAGCGUCU
395
59
||..||||.|.|....||||||
GACUGGCAGAGAAAAAUAGAUG
397
60
||.|.||.|..|.|||.||| |.|||...|||.|.||
CUCUGGAAGACCUGAACACCAGAUGGAAGCUUCUGCA
399
16
||||||||.| ||.||..| |||...||||||.|||
AGAAAAAGCA-AUCCAUGGGCAAACUGUAUUCACUCAA
401
18
||||.||||.||....|.|||.|| |...||||....||||.|.||
CACAGCUGGAUUACUCGCUCAGAA-GCUGUGUUGCAGAGUCCUGAA
403
20
||.|.|||| |||...||||||..|||.| ||||||
AAUGCAGAUAGCAUCAAACAAGCCUCAGAACAACUG
405
23
||.|.||...|||.|||||...|||.|
AAUUAGCCGGAAAUAUCAAUCAGAAUU
407
24
|||..||||||.||.|| |||||.|.|
UUCUAAAAAAGCAGCUGAAACAGUGCAGA
409
25
|||||||...||...||||||.||...||.. ||||..|.| ||..|||...||...|.....|||||.|| ||||||
UUUUAGUCAGUGAUAUUCAGACAAUUCAGCC-CAGUCUAAACAGUGUCAAUGAAGGUGGGCAGAAGAUAAAGAAUGAA
411
26
|||..||.|||||| |.||.||.||.||..|||
AUGAAUUACAGAAA--GCAGUUGAAGAGAUGAAG
413
27
|.|||||||||....||||||
ACAAAAAGAAGCGAAAGUGAA
415
28
||.||||.|||||..|...|..|.|||..||..|||..|...||..|||.|.|
CAAACAAGUGGCUAAAUGAAGUAGAAUUUAAACUUAAAACCACUGAAAACAUU
417
29
|||.|||......||..||||||.||.|
AGAGGAUAACCCAAAUCAGAUUCGCAUA
419
30
||.||||.||.|..||.||||...|.|.|..|||
UUGCAGACAAGGUGGACGCAGCUCAAAUGCCUCA
421
31
|||||..|||.|.|| ||||||.|.|.|
AAAAUCCAAUCUGAUUUGACAAGUCAUGAGA
423
32
|||..|||.|.|||..|..|.||..|||..||||||
GCCAAUUUUGAGCAGCGUCUACAAGAAAGUAAGAUG
425
33
|.|||||||||.|||.|.|
UACAGAAAAAGCAGACGGA
427
34
||||||.| |||...||||.....|.||.|....||.||.|....||||| |.|||.||||||. |.|||||.|.|.|.||||
CAGAAAGA-AAGCAACAGUUGGAGAAAUGCUUGAAAUUGUCCCGUAAGAUGCGAAAGGAAAUGA----AUGUCUUGACAGAAUGGC
429
35
||.|||||.|||||.||||....|..||..|||||.|
UUGAGAAACAGAAGGUGCACCUGAAGAGUAUCACAGA
431
36
||.|||..|...||||||||||....|||..|| ||||
UGGAUGAAUCAGAGAAAAAGAAACCCCAGCAAA-AAGA
433
37
|||||||....||.||||
UUUAAAGGCAGAACUGAA
435
38
|.|||..|||| |||..|...|..|||||. |||. ||||..|||.||.|.||||.||
UCCAUUCCUUU--GAAGGAAUUGGAGCAGUUUAACUCAGAUAUACAAAAAUUGCUUGAA
437
39
|.|||.||||. |||.|.|.|.||.| |.|.|||.|.|...|||.||| |||||...||||.|.|||||
UGAGAGAAAGCGAGAGGAAAUAAAGA----UAAAACAGCAGCUGUUACAGA-CAAAACAUAAUGCUCUCAAG
439
40
||||||||...|..|..|.||| |||.||..|.|||..|. ||..|||.|.|.|...|.||.|..||.|.|||..||.|
AAAGAAGAAAAAAGGCUCUAGAAAUUUCUCAUCAGUGGUAUC--AGUACAAGAGGCAGGCUGAUGAUCUCCUGAAAUGCUUG
441
41
||.|||||.|||||...|.|| ||||
UUGCAGAAGAAGAAAGAGGAGCUGAA
443
42
|.||||.||||||
GACUUUGAAGAUC
445
44
|.||||.|...|||||..|||||...|||.|.....|.|..||||..||.|...||| |..||. ||.|| |.||||. |||||.||||
UAUUUAAUCAGUGGCUAACAGAAGCUGAACAGUUUCUCAGAAAGACACAAAUUCCUGAGAAUUGGGAACAUGCUAAAUACAAAUGGUAUC
447
46
||||.|||||..|.||.||.|||....||||||.|
AAGAACAAAAGAAUAUCUUGUCAGAAUUUCAAAGA
449
47
|||||.||..||||| |....|||||....||||
AAGAUAAACUUGAAAAUAAGCUCAAGCAGACAAA
451
48
||||..|||....||||| |||||.||
GCUUGAAGACCUUGAAGA-GCAGUUAA
453
49
||||..|||...|||||. ||||.| |.|||||.||.|.|
GAAACUGAAAUAGCAGUU--CAAGCU--AAACAACCGGAUGU
455
51
||.|.||.|.|||...||.||.|||.| ||||||
UGACCUUGAGGAUAUCAACGAGAUGAUCAUCAAG
457
53
|||||||..||..| |||.|||. ||.||.|....||.|||.||....|| ||.|.| ||||||||||| |||..|||||.| |.||||
UUCAGAAUCAGUGGGAUGAAGUACAAGAACACCUUCAGAACCGGAGGCAACAGUUGA--AUGAAAUGUUA--AAGGAUUCAACACAAUGGC
459
55
|||..|||.||||||.|||||...|||
UUUCUUGCCUGGCUUACAGAAGCUGAA
461
57
|||||||..|||| ||||.|
UUCAGAAGCAGAACGAUGUA
463
59
||.||..||||.|||.| |||||
UGACUGGCAGAGAAAAAUAGAUG
465
60
||.||||.||||..||
UCUGAAAGAGAACGUG
467
18
||||||||||| ||||
AAAAGAAAAAGUCAAU
469
20
|||||.||.|||..|...|.|.|.||.|| |.|.|.|||.| |.....||...|||||....|.|||.||.|.||..|||..|||.|
UAAGUGAGAGACUUAACUGGCUGGAGUAU--CAGAACAACAU---CAUCGCUUUCUAUAAUCAGCUACAACAAUUGGAGCAGAUGACAAC
471
22
||.|.||||.|.|.||..|.|.||.|||||
AACUUAGUGUCACCGACUAUGAAAUCAUGG
473
23
||||||.| .|.|.||||| ||.||..||||..|.||..||
AAAAGCUA--GAGGAGCAAA-UGAAUAAACUCCGAAAAAUUC
475
24
||||.|||||. |.|.|||||||.|
ACCCUGAAGAA--AUGGAUGGCUGAA
477
25
||||..||||||..||..||. |||||...|.|....||.|....|.||.||.|||||.|.|
AAGAAUGAAGCAGAGCCAGAG---UUUGCUUCGAGACUUGAGACAGAACUCAAAGAACUUAACA
479
26
|.|.||||||||..| |||.|..|||||||
GAGAAAACUGUAAGC-CUCCAGAAAGAUCU
481
27
||||||..|||.||.||||
UUAAAAAAGGAACUUGAAA
483
28
||.|||||||.|.|..|||.||.||.|.|
GAGAAAGCAAACAAGUGGCUAAAUGAAGU
485
29
||||.|.||||..|||||.||
CAGAUGGCGGAGUCAUGGAUG
487
30
|||||.||||.|||..||
CCCAGGAGACUGAAAAAU
489
31
|||||.|||.||.|| |..||.|||.|
AGAAAUGAAGAAACAUAAUCAGGGGAA
491
32
|||.||||.||||
UUAUUCCAGAAAC
493
33
||||| ||||||| |||||||| ||||
CCAAAGAACUUGAUGAAAGAGUAACAGC
495
34
|.|||.|.|||..||.||||..|||
CAGAUAUGGAAUUGACAAAGAGAUC
497
36
||||..|..|.||||||||.||.||
GGAUGAAUCAGAGAAAAAGAAACCC
499
37
|||.|.||| ||||.|...||..||||| |..|.|| |||||||.|
AAAGGUGGACUCUACACGUGACCAAGCAGCAAACUUGAUGGCAAACCG
501
38
|.|||...|.||||.|..|.|||..|.|||||.||..| ||.||||
ACUCAGAUAUACAAAAAUUGCUUGAACCACUGGAGGCUGAAAUUCAG
503
39
||..|.|||....||||.|.||. |..|||.|||||..|||..|.|..|.||.||
AAAAGAAUUGUUGCAAAGAGGAGACAACUUACAACAAAGAAUCACAGAUGAGAGA
505
40
||||..||.||||||||
AAAAAAGGCUCUAGAAA
507
41
|||||||.|.|||....||||.||
CAGAAGAAGAAAGAGGAGCUGAAU
509
42
||||.|.|||||||
ACUGAAAUCACUCA
511
44
||.||||...|| ||||..|.||||| |||..||
ACAGAAGCUGAA--CAGUUUCUCAGAAAGACACAA
513
46
|||||.|.|.|.|..|||..||||||
AAAAGAGCAGCAACUAAAAGAAAAGC
515
47
|.|||||||||||.|
AUAAACUUGAAAAUA
517
48
||.||||..|.||.||..|.||..||..||||| ||||
AGGAAUCAGUUGGAAAUUUAUAACCAACCAAACCAAGA
519
49
|||..|||..|||.||..||.||.|.|.||||
UUUGUACAAGGAAAAACCAGCCACUCAGCCAG
521
51
|.||||||...||.| |||||.|||..|.|..||..|..|||.|
GGAAACUGCCAUCUC-CAAACUAGAAAUGCCAUCUUCCUUGAUG
523
53
|.||.|.|.||||||||...|.|||
ACAGUAGAUGCAAUCCAAAAGAAAA
525
55
|||||.| |||..|||.|...||..||.|.||..|||||
GAAGACUCCAAGGGAGUAAAAGAGCUGAUGAAACAAUGGC
527
57
|||.||..|..||..|||..|.| |.||| |||||..||.|...|.|.|.||.|.||.|
UUCUCUGCAGGAACUUCUGGUGUGGCUACAGCUGAAAGAUGAUGAAUUAAGCCGGCAGGC
529
59
|||..||.|||||.|.||.. |||.||||...| ||||||..||....|.|.|.|||
GCAGCCCGUGGGCGAUCUCCUCAUUGACUCUCUCCAAGAUCACCUCGAGAAAGUCAAG
531
60
||..|||.||||||.|
CACCAGAUGGAAGCUU
533
18
||.|||..|.|||| |.||||..||.|.|
AUUACUCGCUCAGA-AGCUGUGUUGCAGA
535
20
|||..||||.| |||||.|||||..|||
GAAAAUCCAAC-CCACCACCCCAUCAGA
537
22
||||||.|..||.||||
CCACCAAUGCGCUAUCA
539
23
||.||||.|....||||| ||.|..||..|..|.|...|||||||||||
CAGCACCACUGUGAAAGA-GAUGUCGAAGAAAGCGCCCUCUGAAAUUAG
541
24
||.||||||.||||||...||||
UGGGGAUUCAGAAAUUCUAAAAA
543
25
||||||| ||..|.||||..||.|||..||
AAACAGUGUCAAUGAAGGUGGGCAGAAGAUAA
545
27
||.||||||||..|.|.|.|.|..||.|..|..||.|||
AAAAGGAACUUGAAACUCUAACCACCAACUACCAGUGGC
547
28
||..||.||||.|| |||..|||..||.|..||...|.|..||.|..|||.|.|| |||||...|||
AAAGCAAACAAGUGGCUAAAUGAAGUAGAAUUUAAACUUAAAACCACUGAAAACAUUCCUGGCGGAGC
549
29
||.|||...||| |..|||| |||||||||
UCAUGGAUGAGC-UAAUCAAUGAGGAACUU
551
30
||.|||..|..|||..|.|...|||..||||||
AACAGAGCAUCCAGUCUGCCCAGGAGACUGAAA
553
31
|.|.||||.|..|.||..|||.|.|||
AAGAAACAUAAUCAGGGGAAGGAGGCU
555
32
|||.|.||||||||...|.|..||. ||...|||. |.||||...|.|.||||.|||
CUGCAUUGGAAACAAAGAGUGUGGA-ACAGGAAGUAGUACAGUCACAGCUAAAUCAU
557
33
|.||.||.|.||.|...|||.|.||||||.| |||||.|
AUAAAAGUCUGAGUGAAGUGAAGUCUGAAGU--GGAAAUG
559
34
|.||..|.||.|.|||||||||||.||
AUGCCUAGUAAUUUGGAUUCUGAAGUU
561
35
||||||||.|..|||||||
CACCUCCCGAGCAGAAGAG
563
36
|.|||||||||.|
CAAAAAGAAGACG
565
37
|||..|||| |.||||.|| ||..||||||||||
CAGCAAACU-UGAUGGCAA-----ACCGCGGUGACCAC
567
38
||||..||||||||
UGGAGGCUGAAAUU
569
39
|||||.|||.||..||||.... ||.||..|.||.||..||||
AAAGAGGAGACAACUUACAACAAAGAAUCACAGAUGAGAGAAAG
571
40
||..|.|||||||||..|..||... |.|||||||
CAGUACAAGAGGCAGGCUGAUGAUC-UCCUGAAAU
573
41
|||||||.|.|.| |||.||.|..|.||.|
CAGAUCCAGCUCA-GCAAGCGCUGGCGGGA
575
42
|||.||.||.|..|||.||||..||
AAACGAUGAUGGUGAUGACUGAAGA
577
44
||||..||.|||.|||.|.|||..|||
AAGACACAAAUUCCUGAGAAUUGGGAA
579
46
||.||.||||||.||..||
GAGGAAGCAGAUAACAUUG
581
47
|.|||.|||.| ||.|.|..||.| |||..|.|||||||..||
GCCCAGAAGAGCAAGAUAAACUUG-AAAAUAAGCUCAAGCAGA
583
48
|.||||||.| |||...||||||||
UGCUGUGGUUAUCUCCUAUUAGGAA
585
49
|||| |||||||||||
ACCAGCCACUCAGCCA
587
51
|.||.||.|||..|||||| ||||
AGACUGUUACUCUGGUGAC-ACAA
589
53
||.|||..|.|..||.||||.|| ||||.||
CCUUCAGAACCGGAGGCAACAGUUGAAUGAAA
591
55
||.|||..|||.|.||||
CUACCCGUAAGGAAAGGC
593
57
|.|.|.|||...|||||||||
ACCUUUCUCUGCAGGAACUUC
595
59
|.|.|||.||.|..||. |.|.||...||||||.|||.|.||||
GAGCCCAGAAUGUCACUCGGCUUCUACGAAAGCAGGCUGAGGAGG
597
60
|.||||.|..|..|..||.|||||.|..|...|| |||||
UCUCACCGUAUAACCUCAGCACUCUGGAAGACCUGAACAC
599
43
||||||.|.|..||| ||..||||.||||
GAAGCUCUCUCCCAG----CUUGAUUUCCAAU
601
45
||..|||||||.|||..|. .|.|.|||...||||.. |.||||||..||||.|.|
GAAGCCUGAAUCUGCGGUG--GCAGGAGGUCUGCAAACAGCUGUCAGACAGAAAAAAGA
603
54
|||.|||.||..|||||
UAACAGAGAAUAUCAAU
605
56
||.|||...||||| |||||||||
GACGUUUGGAUAACAUGAACUUCA
607
20
|.|.||||.|.|..|||.....|..||||.||.||.|....|.||||||.|..||..||
AAUGCAGAUAGCAUCAAACAAGCCUCAGAACAACUGAACAGCCGGUGGAUCGAAUUCUG
609
22
||||.|.|| ||.|| |||||.|||..||
UCAGGAGAC--CAUGAGUGCCAUCAGGACA
611
23
|||..||||.|.| |.||||.||.|..|||.||...||.||..|.|||....|||...|| |||||.||.||
AGCCGGAAAUAUCAAUCAGAAUUUGAAGAAAUUGAGGGACGCUGGAAGAAGCUCUCCUCCCAGCUGGUUGAGCA
613
24
||||..| ||..|||||..|.||..||||| ||||
AUCACAUACAAACCCUGAAGAAAUGGAUGGCUGAA
615
25
|||||.|||....|||..|| |.||.|||.| |||..||||
AGAGCCAGAGUUUGCUUCGAGACUUGAGACAGAACUCAAAGA
617
26
||||.|.|| |||.....|..|.||.|||. |||||||||.|||...||||.| |||| |.|..|| ||.|...|||| |||.|.|.||||
GCCAGAAAG- GAGGCCUUGAAGGGAGGUUUGGAGAAAACUGUAAGCCUCCAGAAAGAUCUAUCAGAGAUGCACGAAUGGAUGACACAAGCUGAA
619
28
|||||||.|..||....|..|||.||||
AGCUGAGGAAAUCUCUGAGGUGCUAGAU
621
29
||||.| |||.||..||.|||.|||| |||||||||.|.|
AGAUUC--GCAUAUUGGCACAGACCCU-----AACAGAUGGCGGA
623
30
|||..|..||.||..||.|| |.||.|.|.|...|.| ||||.||...||||.|||
CAUUCAUUGACAAGCAGUUG-GCAGCUUAUAUUGCAGACAAGGUGGACGCAGCUCA
625
31
|.|.|.||..|||||||.|.||
ACAAGUCAUGAGAUCAGUUUAG
627
32
|.||||.|.|||||||.|..||
AAAAAAUUACAAGAUGUCUCCA
629
33
|||||||||.|| |||..||..|||... |..||||..||...|.|||||
AGAAAAAGCAGACGGAAAAUCCCAAAGAA-CUUGAUGAAAGAGUAACAGCU
631
34
||.|.||..||.||.||.|| ||||.|.|...|||.||
CAGAAAGAAAGCAACAGUUGGAGAAAUGCUUGAAAUUG
633
35
|.||||..||| |||.|.|..|...|||.|||...|.|.|||.|..||.|..|..|||...|.|||||.|.|
ACCUGAAGAGUAUCACAGAGGUAGGAGAGGCCUUGAAAACAGUUUUGGGCAAGAAGGAGACGUUGGUGGAAG
635
37
|.|||..|.|.|||.|||.|...|||..||...|..||||
GGUGACCACUGCAGGAAAUUAGUAGAGCCCCAAAUCUCAG
637
38
|||||.|.|.|||||
AGCAGUUUAACUCAG
639
39
||.||| |.||||||..|....|||.|..||| ||||.|||
CACAGAUGAGAGAAAGCGAGAGGAAAUAAAGAUAAAACAGCA
641
40
|.|||||...||.|..|||... |..|.|.|||||.||..||.|||.|.|||..|...||.| |||..|.||.|..||
CUAGAAAUUUCUCAUCAGUGGU-AUCAGUACAAGAGGCAGGCUGAUGAUCUCCUGAAAUGCUUGGAUGACAUUGAAAA
643
41
|||||| ||||||| ||.||||.||.|.|
CAGAUCCAGCUCAGCAAGCGCUGGCGGGAAA
645
42
||||||||..|..|.||||
GUGGAACAACUUCUCAAUG
647
44
||.|.||..|.|||...||||||| ||..|||.| |||.|.||.|.|
CAGAAAGACACAAAUUCCUGAGAA-UUGGGAACA-UGCUAAAUACAA
649
46
||||||...||||.|..|||
AGAAAAGCUUGAGCAAGUCA
651
47
|.|||..|||||.| |||||||. |..|.|.||.||||||.||
AUAAGCCCAGAAGA--GCAAGAUAAACUUGAAAAUAAGCUCAAGC
653
49
|||||.||| |||.|....|.|| |||||.||||..||
AGCAGUUCAAGCUAAACAACCGGAUGUGGAAGAGAUUUUG
655
53
|||.||||||||.||.||..|..||.| |.|||..|.|||
GAAGAAGCUGAGCAGGUCUUAGGACAGGCCAGAGCCAAGC
657
55
|||||.|.|.|.||||||.|....||..|||
AGAAGCUGAAACAACUGCCAAUGUCCUACAG
659
57
||.|..|.|||||||.||||
GCGACUUUCCAGCAGUUCAG
661
59
||||.|||||.|||| |.|||.|||| ||.|||
AACUUCAAGAGGCCA-CGGAUGAGCU--GGACCU
663
60
|.||..||||.|..|||.|||| ||.|.|.|.|||.|
AUAACCUCAGCACUCUGGAAGA--CCUGAACACCAGAU
665
21
||..||.|...|.|||||..| ||||..|||.|..|| |..|||||||||
AAGUCAACCGGCUAUCAGGUCUUCAACCUCAAAUUGAA-CGAUUAAAAAUU
667
50
||.||.||||||||
ACCACUAUUGGAGC
669
52
||.|||||..|..|.|..||..||||..||||.| ||||||.|.|..|||.|| |||| |||.|.|.|.||||.|
CAGUUGGAAGAACUCAUUACCGCUGCCCAAAAUU---UGAAAAACAAGACCAGCA----AUCAAGAGGCUAGAACAAUCA
671
58
||.||.|||||..||||..||.|
ACAGAGCAGCCUUUGGAAGGACU
673
22
|||...|||||||||
CAAUGCGCUAUCAGG
675
23
||..||||...|..|||||||||
UCUCAGCACCACUGUGAAAGAGA
677
24
||||.| ||||||.||..|.....||||||.||
GAUUCAGAAAUUCUAAAAAAGCAGCUGAAACAG
679
25
||.|.|||.||. |||..|...|.||.||||...|.|.||||.||...|.||.||
CAGUCUAAACAGUGUCAAUGAAGGUGGGCAGAAGAUAAAGAAUGAAGCAGAGCCA
681
26
|||||||.||| ||||.|
AGAAAGAUCUAUCAGAGA
683
27
|||||.|.||||.| ||||
CUGAAUGGGAAAUG-CAAG
685
28
|||.|||.||.|||.||
AUUUAAACUUAAAACCA
687
29
|||...|.|||||.||.|||.|||
GAUGCGACAUUCAGAGGAUAACCC
689
30
|||||.|||.|..||..|..|.||...|||.||
CCUCACAUUCAUUGACAAGCAGUUGGCAGCUUA
691
31
||.||...||||||.|| ||.|.|| ||||
AAAUCCAAUCUGAUUUGACAAGUCAUGAGA
693
32
||||||. ||..||..||.|.|.||..| |.|||.||||.|| |..||||....|..|.|.|.||| |||||
UGAAGUGGAAAUGGUGAUAAAGACUGGA-CGUCAGAUUGUAC-AGAAAAAGCAGACGGAAAAUCCC--AAAGA
695
33
GAAAGGACAAGGACCCAUGUUCCUGGAUGCAGACUUUGUGGCCUUUACAAAU
|.||||.|...|.||.|.||.|.|||| ||||||| ||.|| |||| ||.|| ||||.|...||||...||||.||
AGAAAUGCUUGAAAUUGUCCCGUAAGAUGCGAAAGGA-AAUGA---AUGU--CUUGA--CAGAAUGGCUGGCAGCUACAGAU
697
34
|.||||||.| |||...||||..|||||
GAGGCCUUGA-AAACAGUUUUGGGCAAG
699
35
||||..|||.||.||.|.....|||||
UUUGCAGCCAUUUCACACAGAAUUAAG
701
36
|||||||||.|||
CUGAAAGAGGAAG
703
39
|||.|.|||.||...||. |||||.||.||||.|
AAAGAAUCACAGAUGAGA---GAAAGCGAGAGGAAA
705
40
|||||.||.|. |||||||.||
AGCCCAGAGAU-GAAAGGAAAA
707
41
||||...|.|..||||..||.||.|.|....|.||..|||...||..|| ||||||
AAGCGCUGGCGGGAAAUUGAGAGCAAAUUUGCUCAGUUUCGAAGACUCA-ACUUUG
709
42
||||||...|||....|.||||...|.|| ||.|||||....|.|...|.. ||.||||..|.|||..||||||||||
AGCCCUAUUAGAAGUGGAACAACUUCUCAAUGCUCCUGACCUCUGUGCUAA-GGACUUUGAAGAUCUCUUUAAGCAAG
711
44
|.||.|||.||..||||||
AGAAAGACACAAAUUCCUG
713
46
||.||||....||||| |.|..|.||||.||.| ||.|||....|||.|| |..||||
UUUCAAAGAGAUUUAAAUGAAUUUGUUUUAUGGU-UGGAGGAAGCAGAUAACAUUGCUA
715
47
|||||.||| |||.|||
AGCCCAGAA-GAGCAAG
717
48
||.||.|||||||..||
UUGAAGCUCAAAUAAAA
719
49
||||.| |||.|||||
UGGAAG-AGAUUUUGU
721
51
||||.|||||.||||
CCAUCUUCCUUGAUG
723
53
|||..||.|.|.||..||||||..||..|.| ||.|||..||.|.|.|||
AAUGAAAUGUUAAAGGAUUCAACACAAUGGC-UGGAAGCUAAGGAAGAAG
725
55
|||.||..|||||| |.|.|||.|.|||| |||||.|
CCCCUGGACCUGGA---AAAGUUUCUUGCCUGGCUUACAGA
727
57
||||||.|.|.|||||...||..|.||
GAUGAAUUAAGCCGGCAGGCACCUAUU
729
59
|||.|.||| |||.||...|.||.|.|||..|.||||
GGCUUCUACGAAAGCAGGCUGAGGAGGUCAAUACUGA
731
60
|.|||||||||||.|
CUCUGAAAGAGAACG
733
50
|||...|||||.|.|| ||.|.|||
GGGCAAAGCAGCCUGA--CCUAGCUC
735
52
||.||||||...||..|||....|.|.||.||
CAACAAUGCAGGAUUUGGAACAGAGGCGUCCC
737
58
|.|||.||||||.|||
CUCUACCAGGAGCCCA
739
24
|||..||..||.|...|||||||||.||.||
AAUCACAUACAAACCCUGAAGAAAUGGAUGG
741
25
||.|..|.|..|||||.|..|||||.||.|
GAGACUUGAGACAGAACUCAAAGAACUUAA
743
26
|||.||....||.||||.|||.||||||..|..|.|||.. |||...|.|||| ||||||
CUGUAAGCCUCCAGAAAGAUCUAUCAGAGAUGCACGAAUGGAUGACACAAGCUG-AAGAAG
745
27
|||.||||.|...||||.|.||..|..||||
CUCUGCACUAGGCUGAAUGGGAAAUGCAAGA
747
28
|.|||||..|||||||
AAAGCAAACAAGUGGC
749
29
||||||.|| ||.||||...|.||.|....||.||||.|.|.||
AUCAAUGAGGAACUUGAGACAUUUAAUUCUCGUUGGAGGGAACU
751
30
|.|||| ||..|.|.||||.|.|||||
AUAUUGCAGACAAGGUGGACGCAGCUC
753
31
|||||. ||.||||||||..||..||
AUCAGU-UUAGAAGAAAUGAAGAAAC
755
32
|.||..|||||...|.|||||||.|
ACUUGCCUGCAUUGGAAACAAAGAG
757
33
|||..||.|||||| |||....||||.||||.||||
AAAGCAGACGGAAA-AUCCCAAAGAACUUGAUGAAA
759
34
||||||||..|...||.|...| ||..|||||.. |..|.|||.....|||.||.|||..|.|. ||..||||...||.....|..|.|.|...|.||||.||||.
AAGAAAGCAACAGUUGGAGAAAUGCUUGAAAUUGUCCCGUAAGAUGCGAAAGGAAAUGAAUGUCU-UGACAGAAUGGCUGGCAGCUACAGAUAUGGAAUUGACAAAG---//
|||..||||..||||..||..||.|..||||
//AGAUCAGCAGUUGAAGGAAUGCCUAGUAAUU
761
35
|.|||...||||||||...||| |||.|.||.| |||||
CUACUCAAAAAGAGAUUGAGAAACAGAAGGUGCAC-CUGAA
763
36
|||||||||...||.|...||.|. |||...|| ||||..||| |...||||||.| ||..|..|||..||.||
ACAAAGUGGAUCAUUCAGGCUGACACACUUUUGGAUGAAUCAGA-GAAAAAGAAACC-CCAGCAAAAAGAAGACG
765
37
||.|||..|||||||
AUUUCACACAGAAUU
767
38
|||||||.|||||
UUUGAAGGAAUUG
769
39
|||||.||||||...||.||..||||
AAAGCGAGAGGAAAUAAAGAUAAAAC
771
40
|||||.||. |||||...|...||.|...||||.|.|||| |.|.||||||...| ||| ||.||| |||||..||||
GAAAUUUCUCAUCAGUGGUAUCAGUACAAGAGGCAGGCUG----AUGAUCUCCUGAAAUGCUUGGAUGA-CAUUGAAAAAA
773
41
|.||..|||.|.|.|||.||||...||..||.| |||.|.|.||..|.....|.||||| .|.|| |||||..|.|.|.|.|..||||
AAAUUGAUCGGGAAUUGCAGAAGAAGAAAGAGGAGCUGAAUGCAGUGCGUAGGCAAGCUG--AGGGC-UUGUCUGAGGAUGGGGCCGCAA
775
42
||..|||..|.||||.| |||||.|. |||.|..||||||
ACGAUGAUGGUGAUGACUGAAGACAU-GCCUUUGGAAAUU
777
44
||||| |.|||...|.||| |.|..||.|.||||.|||.|...|. |||...|||||||
GAAAU--GGCGGCGUUUUCA-UUAUGAUAUAAAGAUAUUUAAUCAGUGGCUAACAGAAGCU
779
46
||||||.||.||||.|||||...|...|.||.||||
GCUAGAAGAACAAAAGAAUAUCUUGUCAGAAUUUCA
781
47
||||.|.||||.|| |||..|.||||.||..||||.|
GGAAUUCUCAAACA--AUUAAAUGAAACUGGAGGACCC
783
48
|.|||||||||.| |.|....|.|||..||....| ||||.|...|..|...|.||.|.||.||.|||||..|.|||.|.||.|
GGAGAAAUUGAAG--CUCAAAUAAAAGACCUUGGGC-AGCUUGAAAAAAAGCUUGAAGACCUUGAAGAGCAGUUAAAUCAUCUGC
785
49
||||.|||||||.|.|...||||.||
UGUGGAAGAGAUUUUGUCUAAAGGGC
787
51
||||||||..||.|||
UUCUCUGCUUGAUCAA
789
53
|||||.|.|| ||.|||..|...|||.|..|||.||| ||||||||||
GAAAUGUUAA--AGGAUUCAACACAAUGGCUGGAAGCUAAGGAAGAAGCU
791
55
||.|||||||.|.|..||.....|.|..||.||.||....|.||||...||..|..|.....||| |.||.||| ||||..||||.|.||
UUGGAAGAAACUCAUAGAUUACUGCAACAGUUCCCCCUGGACCUGGAAAAGUUUCUUGCCUGGCUUACAGAAGC---UGAAACAACUGCCAA
793
57
|||||||||.||.|
UUCUCUGCAGGAAC
795
59
||.|.|.|.||||..|| |||..| |||..||||.|||....|..|.|.|||||...||.||||
CUGCACUCCGCUGACUG-GCAGAG---AAAAAUAGAUGAGACCCUUGAAAGACUCCAGGAACUUCA
797
60
|.|||||..|| |||||||||
GGAGAAAUUGCGCCUCUGAAA
799
25
||||||| ||||| |...|||...||||||||
AUUCAGACAAUUC----AGCCCAGUCUAAACAGUG
801
26
||.|..||||||||.|..|||.|||.|
UGCACGAAUGGAUGACACAAGCUGAAG
803
27
||||||||.|.|| |||||
UAAAAAAGGAACUUGAAAC
805
28
||.||.||.|.|||||.|.||...||||.|| |.|||.|...| |||||.|
ACUUAAAACCACUGAAAACAUUCCUGGCGGA-GCUGAGGAAAUCUCUGAGG
807
29
|||.||||| ||.|.||||..|..|.|.| ||||...|..|.||.||||||
ACAGACCCU-AACAGAUGGCGGAGUCAUG--GAUGAGCUAAUCAAUGAGGAA
809
30
|||||..|||..|||||.|||
AAAAGUUGCUUGAACAGAGCA
811
31
||.||.|..||||||||
UGUCUCAGAUUGAUGUU
813
32
|||||..|.|.|.||||||..| |||..||.|.|.|.|..||.||.|..|...|||||
UGAAGAUGCACUUGCCUGCAUU---GGAAACAAAGAGUGUGGAACAGGAAGUAGUACAGU
815
33
|.|.||.|.|||.|..||..|||||| ||.|| ||||||..|.|||||
AACUUGUAUAAAAGUCUGAGUGAAGU-GAAGU----CUGAAGUGGAAAUGG
817
34
|||||||..||...|...|||...|||.|...|||.| |||||.||||..|.|||
GAAUGGCUGGCAGCUACAGAUAUGGAAUUGACAAAGAGAUCAGCAGUUGAAGGAAUGC
819
35
|||||..||.|| ||.|..|||.|..|.||. ||.|.||||..|||||
AGAAACAGAAGG-UGCACCUGAAGAGUAUCACAGAGGUAGGAGAGGCCU
821
36
||.|||||..||||
AAACACAUGGAAAC
823
38
|||.|.|...|.||||..|.|||| |||||||.||.|. ||...||||||.||||||
ACAAAAAUUGCUUGAACCACUGGA-GGCUGAAAUUCAGCAGGGGGUGAAUCUGAAAGAGGAA
825
39
|||||.||||||||..||||
UAAAACAGCAGCUGUUACAG
827
40
||||||..|||.|||..||||
GAAAUGCUUGGAUGACAUUGA
829
41
|||.||.|||||||. ||||||
AAAGAGGAGCUGAAUGCAGUGC
831
42
||.||...||.||||||||.| |||||
CACACUGUCCGUGAAGAAACGAUGAUGG
833
44
||.|.|||.|.|...|.||..|.|||.|..|.||||.|||. ||||.||.|||.||
AUGAUAUAAAGAUAUUUAAUCAGUGGCUAACAGAAGCUGAACAGUUUCUCAGAAAGA
835
46
||||.|||..||||..|||
AUGGUUGGAGGAAGCAGAU
837
47
||.|||..|||.||.|.||| |||.|..|||.||||..|||...|.||| ||.|.|..|.|.|.||||
UUACUGGUGGAAGAGUUGCCCCUGCGCCAGGGAAUUCUCAAACAAUUAAAUGAAACUGGAGGACCCGUGC
839
48
|||...||..||.|||||.|.||.||
GAAGACCUUGAAGAGCAGUUAAAUCA
841
49
||.||.||..||||| ||.|...||| |||.|||.||||
CUAAACAACCGGAUG--UGGAAGAGAU-UUUGUCUAAAGG
843
51
||.|||||.|||| ||| ||.||..|.|..||||||
UGACCUUGAGGAUAUCAACGAGAUGAUCAUCAAGCAG
845
53
||..||| |.||..|||..||||..||| |||...|||||| ||...|..|.|||||
AAGUACA-AGAACACCUUCAGAACCGGA-GGCAACAGUUGAAUGAAAUGUUAAAGGA
847
55
|||||.|||. |||.|..||.||..|.....|||||.|||.|.|||||||.||
AGGAAAGGCU---CCUAGAAGACUCCAAGGGAGUAAAAGAGCUGAUGAAACAAUG
849
57
||||||.. |.||||..|.||||.|.|.....|.|||.|| ||||......|| ||||||||.|
UUCUGACCAGUGGAAGCGUCUGCACCUUUCUCUGCAGGAACUUCUGGUGUGGCUACAGCUGAAAGA
851
59
|||||..||||||| ||||
UUCUACGAAAGCAGGCUGA
853
26
|||||||.|||..|. |||..|||.|||| ||..|.||.|||
CUUGAGAGAGAUUUU---GAAUAUAAAACUCCAGAUGAAUUACA
855
27
||.||||||.|.||||
AAGGAACUUGAAACUC
857
29
|.|.||||.|.|||.| ||.|||. |.|||.||||
AAAAUUUGAUGCGACA-UUCAGAGGAUAACCCAAA
859
30
||.||.||.||....|....|||.|||.|.|.|.|||||.|.|
GCAUCCAGUCUGCCCAGGAGACUGAAAAAUCCUUACACUUAAU
861
31
|.||.||| ||||.|.||..|||.||| ||||.||.|
AGUUUAGA-AGAAAUGAAGAAACAUAA---UCAGGGGAA
863
32
|||||. |.||..|||||.|.|.|.|.|||.|||.|. |||...|..|||.|||.....||||||
UAUUCC-AGAAACCAGCCAAUUUUGAGCAGCGUCUAC-AAGAAAGUAAGAUGAUUUUAGAUGAAG
865
33
|||||..|....|..||||||||.||.|. |.|||| |||....||.|....||||.||| |.||||||||||.|
UGAAGUGGAAAUGGUGAUAAAGACUGGAC----GUCAGA--UUGUACAGAAAAAGCAGACGGAAAAUCCCAAAGAACUUGA
867
34
||.|.||||||.|.|.|||
AACAGAAAGAAAGCAACAG
869
35
|.||.||..|||||||||
ACGUUGGUGGAAGAUAAA
871
36
|.|||||.|||||..|.||
GGAUGAAUCAGAGAAAAAG
873
37
||..|||.|.|||.||.|||| |.|||..|.||.|.||..||
CAUCGAUUUGCAGCCAUUUCA-CACAGAAUUAAGACUGGAAA
875
38
||.|..||||||..|. |||.|||.|.|||.|
CAAAAAUUGCUUGAACCACUGGAGGCUGAAAU
877
39
||..|||||||. ||...|.|.|||| ||..|...|| ||.|.||..|.||..|| ||.||.|||.||.||
AUGAAGACAAUG-AGGGUACUGUAAA-AGAAUUGUUGCAAAGAGGAGACAACUUACAACAAAGAAUCACAG
879
40
|||.|.|| |||.|..||.||..|| |||||||.||||
AGAAAAAAGGCUCUAGAAAUUUCUCA-UCAGUGGUAUCA
881
41
|.||.|||.||| |||.|..||.||||.|..| ||..||||
GGGAAAUUGAGAGCAAAUUUGCUCAGUUUCGA-AGACUCAA
883
42
|||.||||..|||||....|..||.|.|.|.||.|||||
ACACUGUCCGUGAAGAAACGAUGAUGGUGAUGACUGAAG
885
44
|.||||.|...|.|||.|. ||....||||||.|.||.|.|....|||| |.||.|..||.|| ||||.|||.||
UAUAAAGAUAUUUAAUCAGUGGCUAACAGAAGCUGAACAGUUUCUCAGAAAGACACAAAUUCCU--GAGAAUUGGGA
887
46
|.|||||.| |||||| |.||||
GUCAGAAUUUCAAAGAGAUUUAA
889
47
|||...|.||| |||||.| |.||||||..|||
CAGGGAAUUCUCAAACAAU-UAAAUGAAACUGG
891
48
|||||.|.|.|.||..||||.|||.|....||||...||||..||
CUUGAAAAAAAGCUUGAAGACCUUGAAGAGCAGUUAAAUCAUCUG
893
49
|||..||..|||||||
CAGUUCAAGCUAAACA
895
51
||.||.|..|..|.|||| |||||||||.|..||
AGAUUUCAACCGGGCUUG-GACAGAACUUACCGA
897
53
|.|||||| ||.|.|.||||||||||.|
UGAAAGAA-UUCAGAAUCAGUGGGAUGA
899
55
|.|||||..|.|..|.||| |||||| ||.|||.||||
AAAGUUUCUUGCCUGGCUU---ACAGAAGCUGAAACAACU
901
57
|||||.|.||||.||||.|
AAGAUGAUGAAUUAAGCCG
903
59
|||||| |||.|.|...|||.....|..||.||.|..|..||.|..| ||.|||.|||||||..|..||..||..||
AGAGCCCAGAAUGUCACUCGGCUUCUACGAAAGCAGGCUGAGGAGGUCAAUACUGAGUGGGAAAAAUUGAACCUGCA
905
27
||||.||||||||...|...|.|.|||... |||..|.|||||||..| |||.| |.|.||..|||...|.||.|.|||
AGCUAAAGAAGAGGCCCAACAAAAAGAAGC-GAAAGUGAAACUCCUUACUGAGU--CUGUAAAUAGUGUCAUAGCUCAAG
907
28
|||...|||.|||.||..||||||..|.||..|..|.|.....|.||.|||.||.||
UGAAGUAGAAUUUAAACUUAAAACCACUGAAAACAUUCCUGGCGGAGCUGAGGAAAU
909
29
|||.|||| |..|.|...|||||||| |||.|..|| |||.|.|||||||.|
CUAACAGAUGGCGGAGUCAUGGAUGA-----GCUAAUCAAUGAGGAACUUGAGACA
911
31
||.|.||||| |||||.||||||
GAGAUCAGUUUAGAAGAAAUGAAG
913
32
|||.|||||||..||.|..|...|.|..|..|..|.||.|..|||||. |......||||..|....||||||||
UAUUCCAGAAACCAGCCAAUUUUGAGCAGCGUCUACAAGAAAGUAAGA-UGAUUUUAGAUGAAGUGAAGAUGCAC
915
33
|||||||.||....||||.|..||...|.|||.|||..|.|||
UAUAAAAGUCUGAGUGAAGUGAAGUCUGAAGUGGAAAUGGUGA
917
34
|||.||||..|||....|..|..|||.|| |||.| ||.|.||||||.|||.....||.. ||.|.|..|..|..|||...|.|||. |||||||||||..|||
AGAAAUGCUUGAAAUUGUCCCGUAAGAUGCGAAAGGAAAUGAAUGUCUUGACAGAAUGGCUGGCAGCUACAGAUAUGGAAUUGACAAAGAGAUCAGCAGUUGAAGGAAUG
919
35
|||||||||||...||.|||||..||.|..|..|...|...|.|||||..| ||||
GAGGCCUUGAAAACAGUUUUGGGCAAGAAGGAGACGUUGGUGGAAGAUAAACUCAG
921
36
|.||.|..|||.||..|.|.|||...||.| ||.|||| |||..||||. ||..|||...|||||||..|| ||...|||||
AUGGAAACUUUUGACCAGAAUGUGGACCACAUCACAAAGUGGAUCAUUCAGGCUGACACACUUUUGGAUGAAUCAGAGAAAAAGAA
923
37
|.|.|||..|.||.||||||.|.||..||
AAAGGCAGAACUGAAUGACAUACGCCCAA
925
38
|||||..|.||..|||.|.|...||.|...||.|..||...||...||.|.| |||||.|.|||.. .||.||.||..|.||||
UCAGAUAUACAAAAAUUGCUUGAACCACUGGAGGCUGAAAUUCAGCAGGGGG----UGAAUCUGAAAGA--GGAAGACUUCAAUAAAG
927
39
|.|||||....|.||.|||||||..|||.|| |||||||.|
GCGAGAGGAAAUAAAGAUAAAACAGCAGCUG--UUACAGACA
929
40
|||||.|..|..||.....|||.|||.|||||..||| |||||.|.| |..|.|..|.||..||.|||||.||
GAGGUCUCAAAGAAGAAAAAAGGCUCUAGAAAUUUCUCAUCAGUGGU--AUCAGUACAAGAGGCAGGCUGAUGA
931
41
|||||.|||||.......||.|||.||||
GAAUUGCAGAAGAAGAAAGAGGAGCUGAA
933
42
||||.|||...||.|||||
UUUGAAGAUCUCUUUAAGC
935
44
||.|.||..||.||.||...||.|. ||..|.|||.||. |||.||..|...| |||.|.|||.||||||||.| ||.|||. |||.|||
AGAUCUGUUGAGAAAUGGCGGCGUUUUCAUUAUGAUAUAA-AGAUAUUUAAUCAGUGGCUAACAGAAGCUGAACA---GUUUCUC-AGAAAGA
937
46
|.||||.|||||||..||| ||||..||.|.| |..|.|||||||
AAAAGAAUAUCUUGUCAGA-AUUUCAAAGAGA----UUUAAAUGAAUU
939
47
||.|..|.||..||..||||..|.|||||.|. ||.|...|.|.|.|||.||||||..||
AUAAGCCCAGAAGAGCAAGAUAAACUUGAAAAUAAGCUCAAGCAGACAAAUCUCCAGUGGA
941
48
|||||| |.|.||.|.||||..||.|.|...|| ..|||||.||..|.|..|.||||.|. |..||||| ||||..|..||.||..|..| |.|||||||.||
UCCAGA--GCUUUACCUGAGAAACAAGGAGAAAU---UGAAGCUCAAAUAAAAGACCUUGGGC---AGCUUGAA---AAAAAGCUUGAAGACCUUGA-AGAGCAGUUAAA
943
49
||||||.||.||..|.||.|.|| ||||||||||..||.|...|..||..||.|..|. |.|||.|||
CAAGCUAAACAACCGGAUGUGGA-AGAGAUUUUGUCUAAAGGGCAGCAUUUGUACAAGGAAAAACCAG
945
51
||.|||.|.|.|||.|..|.|| |.|||||| ||.||.....|.||.|...||.||..|||
AUCACAGAGGGUGAUGGUGGGUGACCUUGAG-GAUAUCAACGAGAUGAUCAUCAAGCAGAA
947
53
|||| ||.|| |||.||....|||....||..|.|||||||| ||.|||
UGAAGUACAAGAACACCUUCAGAACCGGAGGCAACAGUUGAAUGAAAUG
949
55
||.|||..||.|||.|.|.|..|... |||..||||.||...| ||.....||.||...|.||.|.|..|..|||.|.|||.||||||||
GAGAGGCUGCUUUGGAAGAAACUCAU-AGAUUACUGCAACAGUUCCCCCUGGACCUGGAAAAGUUUCUUGCCUGGCUUACAGAAGCUGAA
951
57
||||||||| ||...||||
GAUGAAUUA-AGCCGGCAG
953
59
||.|||||.||.|...||..||||.|.||.. |||||..|..| |||| |.|||.|..||||.|..|.|.||.||||
CAGGCUGAGGAGGUCAAUACUGAGUGGGAAAAAUUGAACCUGCA-CUCC-GCUGACUGGCAGAGAAAAAUAGAUGAGA
955
28
|||||.||| |.||..||| |||.||.||..|..||||||
ACAAGUGGC-UAAAUGAAGUAGAAUUUAAACUUAAAACCAC
957
29
|.||..||||||||||.||
UCAAUGAGGAACUUGAGAC
959
30
|||||||...||.|..|.|||.||
AGCUCAAAUGCCUCAGGAAGCCCA
961
31
|||.||| ||||...||.|.....|..|||.|||...|.||.|||.|..|||| |.|..| |||.|||||
AAAUGAA--GAAACAUAAUCAGGGGAAGGAGGCUGCCCAAAGAGUCCUGUCUCA-GAUUGA--UGUUGCACA
963
32
||||....||||||.|.|||.||.|||||.. ||||.|.|||||
AGUGAAGAUGCACUUGCCUGCAUUGGAAACA-AAGAGUGUGGAA
965
33
||.||||.|||..|.|| ||..|..||....|||| |.|.|||..|.|..|||| ||.||||||||
UGGAAAUGGUGAUAAAGACUGGACGUCAGAUUGUACAGAAAAAGCAGACGGAAAAUCCCAAAGAACUUGA
967
34
||||.||||||| ||||..||.|.||.||.|
AAGAUGCGAAAGGAAAUGAAUGUCUUGACAGAAU
969
35
|||.|||||. |.||||||..| ||||||.. |.||.||.| ||||.|||||...|||||
ACUCAGUCUUCUGAAUAGUAACUGGAUAGCUGU--CACCUCCCG-AGCAGAAGAGUGGUUAAA
971
36
||||..||||||. .||||.||||||||||
AGAGAAAAAGAAA--CCCCAGCAAAAAGAAG
973
37
|.|.|||.|||||||.| ||.|..||.|||
AAUCUCAGAGCUCAACCAUCGAUUUGCAGC
975
38
||.|||||..||||. |.|||||||
GGGUGAAUCUGAAAGAGGAAGACUU
977
39
|||||.|||.| |..|||| ||||||.|..|..|||.|
AAAGAGGAGACAACUUACAACAAAGAAUCACAGAUGAGA
979
40
||||||||..||.||||.| ||.|.||
AAAAAAGGCUCUAGAAAUUUCUCAUCA
981
41
|||..|||||.||||
UGAUCGGGAAUUGCA
983
42
||||.|.|||| ||.|.||...|.|..|.|.| ||.||....|.|.|||..||||||.....|||.||..|||||
GAAAUUUCUUA-UGUGCCUUCUACUUAUUUGA---CUGAAAUCACUCAUGUCUCACAAGCCCUAUUAGAAGUGGAAC
985
44
|.|.|||..||||.| ||.|..|||||..||
UAUUUAAUCAGUGGC-UAACAGAAGCUGAAC
987
46
|.||..|.|||||...||||.|||||||..|...|.|.||.||
ACCUGGAAAAGAGCAGCAACUAAAAGAAAAGCUUGAGCAAGUC
989
47
|||||.|.|||..|||||
AGAAGAGCAAGAUAAACU
991
48
||||||..|...|||...|||..|.|| |||||..||.|.|||..||||....||..||.|||.|
AGAGCUUUACCUGAGAAACAAGGAGAAAUUGAAGCUCAAAUAAAAGACCUUGGGCAGCUUGAAAAAA
993
49
|.||.||..||..|..|||...|||.||.||.|.|||.||
AACUGAAAUAGCAGUUCAAGCUAAACAACCGGAUGUGGAA
995
51
|||..||.|.||||...|.|||.||.|...||.|.|||
UUACUAAGGAAACUGCCAUCUCCAAACUAGAAAUGCCA
997
53
||||||.|||||.||..|.||
AGCUAAGGAAGAAGCUGAGCA
999
55
|.|||||.|||.||.| ||.||..|.||| |...|.||.|.|||.||
AACAACUGCCAAUGUC-CUACAGGAUGCU--ACCCGUAAGGAAAGGCU
1001
57
||||||||. |||||||
ACCAGUGGAAGCGUCUGCAC
1003
59
|||||||..|||||.|||.|.|
CUGCACUCCGCUGACUGGCAGA
1005
60
|||.|||.||||..|.|.|.|.|||
AAGAGAACGUGAGCCACGUCAAUGA
1007
29
|||..||||.|.|.|.|.|.|. ||||..||||..|.|.|.|.|..|..|||.|....|...|||..|.|||||
AUUCGCAUAUUGGCACAGACCCUAACAGAUGGCGGAGUCAUGGAUGAGCUAAUCAAUGAGGAACUUGAGACAUU
1009
30
|||||||.....||||| ||.||..||.|.|.|||.. |||.||.||.|
GUUGGCAGCUUAUAUUG-CAGACAAGGUGGACGCAGCUCAAAUGCCUCA
1011
31
||||||||.|..||
AAAUGAAGAAACAU
1013
32
||||||.||. |.|....||.||||||| |||..|..||||.....||.||.|||||
AGAAAGUAAG-AUGAUUUUAGAUGAAGU-GAAGAUGCACUUGCCUGCAUUGGAAACA
1015
33
|.||..|.||.|.||||||..|.|.|.||.|..|||
AUAAAAGUCUGAGUGAAGUGAAGUCUGAAGUGGAAA
1017
34
||||||||| ||..|||..|||||
AGAAAGCAA-CAGUUGGAGAAAUG
1019
35
||||.|..|||||.|||||
AGCAGAAGAGUGGUUAAAU
1021
36
||||||...||......||.|||.||||..|...||||
GAGAAAAAGAAACCCCAGCAAAAAGAAGACGUGCUUAA
1023
37
||||||.|.|||..|||
AGAAUUAAGACUGGAAA
1025
38
||.||.||.|.|||.||.||
CUGAAAGAGGAAGACUUCAA
1027
39
|||.||.|| ||.||..|.||...|.||..|||..|.||.|||.||
AAAGAAUUGUUGCAAAGAGGAGACAACUUACAACAAAGAAUCACAGA
1029
40
|.||.||.||||.|.|| |..|||.||...|.||.||..|.||.|.|..|.||. |||||.||..|| ||||..||||.|
GUCUCAAAGAAGAAAAAAGGCUCUAGAAAUUUCUCAUCAGUGGUAUCAGUACAAGAGGCAGGCUGAUGAUCUC-CUGAAAUGCUUG
1031
41
|||||..||.|.| |.|...||||||||...|.|||||||
CUGAAUGCAGUGCGUAGGCAAGCUGAGGGCUUGUCUGAGG
1033
42
||..||||||.|||.|.||||
CUUUGGAAAUUUCUUAUGUGC
1035
44
|||..|..|||..|||.|.. |.|||.|.||| |..|||.|...||..|||||||||..||||..|||......||.|.||..||..|| |||||||
GUUGAGAAAUGGCGGCGUUU-UCAUUAUGAUA--UAAAGAUAUUUAAUCAGUGGCUAACAGAAGCUGAACAGUUUCUCAGAAAGACACAA---AUUCCUG
1037
46
||||.||..|.|.||..||||||.|||||.|.||. |.||||
AUGAAUUUGUUUUAUGGUUGGAGGAAGCAGAUAACAUUGCUA
1039
47
||||..|.|||.| |.|.||.||.|.|||..|||.||.||
AAGCCCAGAAGAG-CAAGAUAAACUUGAAAAUAAGCUCAA
1041
48
||..|||.||||.||.||..|.|||. ||||.| |||.|.|.|||| ||..|||....|||.|..||.||
AAACAAGGAGAAAUUGAAGCUCAAAU-----AAAAGA--CCUUGGGCAGCUUGAAAAAAAGCUUGAAGACCUUGA
1043
49
||.||||.|||.|.||.||..||||
AACUGAAAUAGCAGUUCAAGCUAAA
1045
51
||||||.|.|.|..||. |.|||||...|....|.|| |.||..| |||||.|||.||......|..|||..||.|| |||||.|||...|||.||.|
CUUGGACAGAACUUACCGACUGGCUUUCUCUGCUUGA--UCAAGUUAUAAAAUCACAGAGGGUGAUGGUGGGUGACCUUGAGGAUAUCAACGAGAUGAU
1047
53
|||.||||. |||..|.|||..|.|.||.|||.|. |||| .||||.|||||....|||||
AGUUGAAUG-AAAUGUUAAAGGAUUCAACACAAUGGCUGG--AAGCUAAGGAAGAAGCUGAG
1049
55
|||||.|.| |..||||.||...||||....|||.|||...||.|.|||
AAACUCAUAGAUUACUGCAACAGUUCCCCCUGGACCUGGAAAAGUUUCU
1051
57
|||.||.||.|..|||||.|
GCUGAAAGAUGAUGAAUUAA
1053
59
|.|.||||.||.||..|..||.||||
GAAAAAUUGAACCUGCACUCCGCUGA
1055
30
||||||.|....||.|....|.|..||||.|....|||.....|.|.|.||.|.....||.||||.|||
CUUGAACAGAGCAUCCAGUCUGCCCAGGAGACUGAAAAAUCCUUACACUUAAUCCAGGAGUCCCUCACA
1057
31
||||||| |||.||.| ||..|||||.|||
AGUCAUGAGAUCAGUUUAGAAGAAAUGAAGAA
1059
32
|.|.|.|.|.|||..|.|.|...||.||||..||||. ||.|.|||
AAAAUUACAAGAUGUCUCCAUGAAGUUUCGAUUAUUC-CAGAAACC
1061
33
|.||.||||||..|.|.|.|.||
AGAACUUGAUGAAAGAGUAACAG
1063
34
|||||..|.| ||||.||.|||....|.|||.||..|||.|
CAGAUAUGGA-AUUGACAAAGAGAUCAGCAGUUGAAGGAAU
1065
35
||||||.||.|.| |.|| |.||| .||||..||||.|...|||.|.||.||
AACAGAAGGUGCA--CCUG-AAGAG--UAUCACAGAGGUAGGAGAGGCCUUGAA
1067
36
|||.||.||| |||||...|| ||...||..|.|.|.||||...||| |||...|||..|.||.||....||||
CACAUGGAAACUUUUGACCAGA-AUGUGGACCACAUCACAAAGUGGAU---CAUUCAGGCUGACACACUUUUGGAUG
1069
37
|.|.|||.|.|.||.|||.|| |..|..||..|..|||.|||..||
GGAAAUUAGUAGAGCCCCAAAUCUCAGAGCUCAACCAUCGAUUUGC
1071
38
|||||.|.|||...|||||.||
AGGAAUUGGAGCAGUUUAACUC
1073
39
||.|||..||||||..||.....||||||.|..|||.|
GAAUCACAGAUGAGAGAAAGCGAGAGGAAAUAAAGAUA
1075
40
||.|.|||||. ||.||| ||||||.|.|
UCUCCUGAAAUGCUUGGAU--GACAUUGAAA
1077
41
|.||||.||||||.|
AGCCAACUCAGAUCC
1079
42
||||..|||||..|||.|
CACAAGCCCUAUUAGAAG
1081
44
|.||||.||.||||..|| |.|||.|||.||.|..|.||||.|. |||.|.|..|||
AAUCAGUGGCUAACAGAAGCUGAACAGUUUCUCAGAAAGACACAAAUUCCUGAGAAUUGG
1083
46
||..|||.|.|.||.||..|.||||||..|.|.||
GAGCAGCAACUAAAAGAAAAGCUUGAGCAAGUCAA
1085
47
||||||||||..|.| || |||| |.|||.||||||
ACUUGAAAAUAAGCUCAAGCAGACAAAUCUCCAGUGGAUAA
1087
48
|||.|||.||||||
UCAGUUGGAAAUUU
1089
49
||||||.|||.. |||..|.|| ||.||||
AGCUAAACAACC-GGAUGUGGAAGAGAUUU
1091
51
||||.||.| ||| ||..||...||||||||.|.||.|
AACUAGAAAUGCCAUCUUCCUUGAUGUUGGAGGUACCUGC
1093
53
||||.|.|.||.|...||..|||...|||.||||.|||.|
UUGAAAGAAUUCAGAAUCAGUGGGAUGAAGUACAAGAACA
1095
55
||.||||| |||..||| |.|.|.||||.|.|.|..|||.|...|.|..|.||..||.....|||| |||...|.|||
CUGGAAAAGUUUCUUGCCUGGCUUACAGAAGCUGAAACAACUGCCAAUGUCCUACAGGAUGCUACCCGUAAGGAAAGGC
1097
57
|||.||.|||||.|||
AUUGGAGGCGACUUUC
1099
59
|||||.||||..|..||.||.|..||....|. |||..||||..|....||| |.||||.|.|..|..|....|||.| |.|||||||||
GAAAAAUUGAACCUGCACUCCGCUGACUGGCAGAGAAAAAUAGAUGAGACCCUUG--AAAGACUCCAGGAACUUCAAGAGGCCACGGAUGAGCU
1101
31
|||..|||..||.|..||| |..||.||..|||.||||
UAAUCAGGGGAAGGAGGCU-GCCCAAAGAGUCCUGUCU
1103
32
|.|.|||.|||.|.|| ||||.|.||||.|.|
AGAUGCACUUGCCUGC----AUUGGAAACAAAGAG
1105
33
||.||||.|||||..||..|.| |||.||..|.||||..| |.|.|||...|.|.||..||.|||.|||
AGCAGACGGAAAAUCCCAAAGAACUUGAUGAAAGAGUAACAGCUUUGAAAUUGCAUUAUAAUGAGCUGGGAGC
1107
34
|||||| |...|.||||||||. ||||.|.| |||||.|.|..| ||||.|.|| ||||||
UUGACAGAAUGGCUGGCAGCUACAGAUAUGGAAUUGACAAAGAGAUCAGCAGUUGAAGGAAUGCCU
1109
35
|.|||.||| |.|.||||.|.|||.|.|. |..||||||.|..|..|| ||||.|.|
GCAAGAAGG-AGACGUUGGUGGAAGAUAA-ACUCAGUCUUCUGAAUAGUAACUGGAUA
1111
36
|.||.||..|||.|.||| |||...|.||.||||||. ||.|.|||| ||..|||
AGAAACACAUGGAAACUU---UUGACCAGAAUGUGGACCACAUCACAAAGUGGAUCA
1113
37
|||||.||||||||||
CAAAAAUUGCUUGAAC
1115
38
||||...|.||.|||||||
CAGCAGCUGUUACAGACAA
1117
39
||||...|.||.|||||||
CAGCAGCUGUUACAGACAA
1117
40
||||||.||..|.||.|| |||.||
CAUUGAAAAAAAAUUAGCCAGCCUA
1119
41
|||||..||.||||.||.| |||||.||||.|.|
GUGGAGCCAACUCAGAUCCAGCUCAGCAAGCGCUG
1121
42
||..|.|||..||||...||||.|...|||..|...|. |||||||.||.|
CCUCUGUGCUAAGGACUUUGAAGAUCUCUUUAAGCAAG---AGGAGUCUCUGA
1123
44
|.|||||.|||.||.|
CACAAAUUCCUGAGAA
1125
46
||.|||.||||||.|.|.|
CUGGAAAAGAGCAGCAACU
1127
47
||||||.| ||.|| |||..|..||||||.|||...|.|.|..|||..|..||| ||.||.||.|
UGCUUGUA-AGUGC-UCCCAUAAGCCCAGAAGAGCAAGAUAAACUUGAAAAUAAGCUCAAGCAGAC
1129
48
|||...||.|||||||.|..|||||
AUUAGGAAUCAGUUGGAAAUUUAUA
1131
49
|.|||.| ||||..|.||| |..|.|.|.|.||.|||.||.|||
UCUAAAG-GGCAGCAUUUGUACAAGGAAAAACCAGCCACUCAGCC
1133
51
|||||.|||||.|.|.|.|.|||.|
GCUUGGACAGAACUUACCGACUGGC
1135
53
||||||||.|||||..||||
GGAGGCAACAGUUGAAUGAA
1137
55
|.|||||||.||||
GGAAAAGUUUCUUG
1139
57
|||||||||.|.|
AAAAAUCCUGAGA
1141
59
|||.||..|.||||.|.|.||||
UGAUCAAGGGAUCCUGGCAGCCC
1143
60
|||..|.|||.|||.|||||.|
UGAACACCAGAUGGAAGCUUCU
1145
32
|||||..| |.||||.|.||||||.|||.||||||..||
ACAAGAAAGUAAGAUGAUUUUAGAUGAAGUGAAGAUGCA
1147
33
|||.|..||..||||.|| |.|..|||.|.|||...|.||||. .||.||...||...|||||||
AAGACUGGACGUCAGAUUGUACAGAAAAAGCAGACGGAAAAUCC---CAAAGAACUUGAUGAAAGAGU
1149
34
||.|||||| |.||||||||.. |.||||.||||
GAAUUGACA----AAGAGAUCAGCAGUUGAAGGAAUG
1151
35
|.||||.||..|||||||.||
AAAAGAGAUUGAGAAACAGAA
1153
36
|||.||.|||....|||||.|.||.|
UUUGGAUGAAUCAGAGAAAAAGAAAC
1155
37
|||.|.|...||||.|.| ||.||.||.|||..||
CAAACCGCGGUGACCACUGCAGGAAAUUAGUAGAG
1157
38
||||.|| .|||| |||..||..||.|||||||....|.||..|.|||.||
UCCAUUC--CUUUG--AAGGAAUUGGAGCAGUUUAACUCAGAUAUACAAAAAU
1159
39
||||||..|....|||.|||||.||||.|.|..|.|||
AUGAGAGAAAGCGAGAGGAAAUAAAGAUAAAACAGCAG
1161
40
||.|||.|| |.||.||.||.|| |||.|||.||.|.|
UCCUGAAAU--GCUUGGAUGACAUUGAAAAAAAAUUAGC
1163
41
|.||||.|....||..|.|||||||.|||||
AAUUGAGAGCAAAUUUGCUCAGUUUCGAAGA
1165
42
|||||||.||.||
GAAGAAACGAUGA
1167
44
|.||.||.||||.|..|||||||.|| |..||.||||..||
AUGAUAUAAAGAUAUUUAAUCAGUGGCUAACAGAAGCUGAACA
1169
46
||||||||...|||||
UAGAAGAACAAAAGAA
1171
47
||||..|...||.||...||||||.....|.||||.| |||.||...|||.||.|
AAGUGCUCCCAUAAGCCCAGAAGAGCAAGAUAAACUUGAAAAUAAGCUCAAGCAGAC
1173
48
||||..||.||.|..|.|.|...|.||.| |||.||..||.||| ||||||
AAAUUGAAGCUCAAAUAAAAGACCUUGGG--CAGCUUGAAAAAAAGCUUGAAGA
1175
49
|.||||| ||...||.|.||||.|
GUGGAAGAGAUUUUGUCUAAAGGG
1177
51
||||.||...|||||.||.|.||||
UCUGCUUGAUCAAGUUAUAAAAUCA
1179
53
|||||||||.|.||.|.|.|||
GGAAGGAGGGUCCCUAUACAGU
1181
55
||.||||..|||.||||||
UAAAAGAGCUGAUGAAACA
1183
57
|||.||...|.|.|...|.|||||.|||||.|
AUUGGAGGCGACUUUCCAGCAGUUCAGAAGCA
1185
59
|.||||.||||....|||.||||
UGAUCAAGGGAUCCUGGCAGCCC
1187
60
|.||.|.|.|||||..||||.|
AGCACUCUGGAAGACCUGAACA
1189
33
||||||||||....|..|| |.|.|||..|.|||||.||. ||...|..|.||||||..|.|||
UGAAGUGAAGUCUGAAGUG---GAAAUGGUGAUAAAGACUGG---ACGUCAGAUUGUACAGAAAAAGC
1191
34
|||||||| ..|.|.|||.||||..| ||||.|.|.|| |||.|.|.|....|.|||||
GUAAGAUG---CGAAAGGAAAUGAAUGU---CUUGACAGAAUGGCUGGCAGCUACAGAUAUGGAA
1193
35
|||.|.|||.||.|.|||||.|.||
AAAAAGAGAUUGAGAAACAGAAGGU
1195
36
||.|||||||| ||...|| ||||||.|||..|.|..|..|.|..|..|.|....|...|.|.|...|.|..|..|||..||.||..||.|.|||||
AUACCAGAAACACAUGGAAACUUUUGACCAGAAUGUGGACCACAUCACAAAGUGGAUCAUUCAGGCUGACACACUUUUGGAUGAAUCAGAGAAAAAGA
1197
37
|||.||||.||.|||.|
UCAGAGCUCAACCAUCG
1199
38
||.|.||.||.||||||
GAAUCUGAAAGAGGAAG
1201
39
|||||.||||| |..|||||.|..|..|||||.||.|.|..|||
GGAAAUAAAGA---UAAAACAGCAGCUGUUACAGACAAAACAUAAU
1203
40
|||||||. |.|.|.|||| |||....|||| |||| |||..|| |||||.|||.|||
UACAAGAG-GCAGGCUGAU----GAUCUCCUGAA-AUGC--UUGGAUGACAUUGAAAAAAAA
1205
41
|.||||.|.|..||||..|.|||.|.|.|.|.|||.| ||.|| ||||.|....||||.||
GAAUUGCAGAAGAAGAAAGAGGAGCUGAAUGCAGUGCGUAGGCA-AGCUGAGGGCUUGUCUG
1207
42
|||.|| ||||||..| |||||. |||||| |.||||| |||||||
AAGAAACGAUGAUGGU-GAUGAC-UGAAGA----CAUGCCU---UUGGAAA
1209
44
||||..|||||...|.|||. |..|||.|.|||.|.|||..||||..| ||..|.|||....||| |||..||..|.||||. |||||.||||
GUUUUCAUUAUGAUAUAAAG-----AUAUUUAAUCAGUGGCUAACAGAAGCU--GAACAGUUUCUCAGAA----AGACACAAAUUCCUGAGAAUUGGGAACA
1211
46
||.|..||.|.|.|||.|||.. ||||.|..||. |||||||..|||.||||||
AUAACAUUGCUAGUAUCCCACUUGAACCUGGAAAA---GAGCAGCAACUAAAAGAAA
1213
47
|||||...|..||..||||.||||.|.|
GAGCAAGAUAAACUUGAAAAUAAGCUCA
1215
48
|||||..|.||..|.||.| |.|.||.|.|| |..|..|.|.|.|| |.||||.||||..||..||.|..|||| || |||.||||||||
AGAAACAAGGAGAAAUUGAAGCUCAAAUAAA-AGACCUUGGGCAGC--UUGAAAAAAAGCUUGAAGACCUUGAAG-AG--------CAGUUAAAUCAU
1217
49
|.||||||||||.|
AAAAACCAGCCACU
1219
51
||||.||...|.||.||..|...|.|.||||..|.|.|||..| ||.|||
UUGAUCAAGUUAUAAAAUCACAGAGGGUGAUGGUGGGUGACCUUGAGGAU
1221
53
|||||.|.||| ||. |..||.||||...|||.||||.| |.|||.||
UGGAAGCUAAGGAAGAAGCUGAGCAGGUCUUAGGACAGGCCAGAGCCAA
1223
55
|.|||||.| ||||||.|....|||..| ||||..|.|||.|| |.|.|..||||.||
CUGAAACAACUGCCAAUGUCCUACAGGAUGCUACCCGUAAGGAA-AGGCUCCUAGAAGA
1225
57
|||||.||.|||.|||.|
AGCCAGUUCUGACCAGUG
1227
59
||..||.||||..||||||...|||..|.|.|.|.|||.|
GAAAAAUUGAACCUGCACUCCGCUGACUGGCAGAGAAAAA
1229
60
||..|.|.|||| ||.|..||||.|.|..|||.|..||
AGGAGAAAUUGC--GCCUCUGAAAGAGAACGUGAGCCAC
1231
34
|.|||.|..||.|.|| |||.|..||.|.||||||||.|
AGUUGAAGGAAUGCCU-AGUAAUUUGGAUUCUGAAGUUG
1233
35
|.||..|||||..|||| ||||.||.|...||.....||..|.||.|.| |||.|.||||
ACUCAAAAAGAGAUUGA-GAAACAGAAGGUGCACCUGAAGAGUAUCACA--GAGGUAGGAG
1235
38
|||||..||..||||..||||| ||||.|..|||||
AGCAGGGGGUGAAUCUGAAAGAGGAAGACUUCAAUAAAGA
1237
39
|||||..|..||||..|.|..||.|.||.......||.||||||...|||||| ||..|||.|..||||||.|||.||.|
AACUUACAACAAAGAAUCACAGAUGAGAGAAAGCGAGAGGAAAUAAAGAUAAA------ACAGCAGCUGUUACAGACAAAACAUA
1239
40
||..|||.||||.|| |..||||. ||...||.|.|.|...|.|||| |.||| |||.|.||| |||.|.||..|||.| |||....|.|||||.|..|.||.|
GAUUUGAGGUCUCAAAGAAGAAAAAAGGCUCUAGAAAUUUCUCAUCAG-UGGUAUCAGUACAAGAGGCAGGCUGAUGAUCUCCUGAAAUGCUUGGAUGACAUUGAAAAA
1241
41
||||||. |..|||.||..||.|.|||.||||
UCAGAUCCAGCUCAGCAAGCGCUGGCGGGAAAU
1243
42
|||||.....|||.||.|.|.|||||.|
UGAAGAAACGAUGAUGGUGAUGACUGAA
1245
44
||||| |.||.||.|..|..||||||
AAAGAUAUUUAAUCAGUGGCUAACAG
1247
46
||||...|.|||||..||||. ||||.|||
AACUAAAAGAAAAGCUUGAGC-AAGUCAAG
1249
47
||.||.||.||||.||.|..|||||| |||.|.|.|.. |||.||.|||
GGGAAUUCUCAAACAAUUAAAUGAAACUGGAGGACCCGUGCUUGUAAGUGC
1251
48
||||.|...||||.|||||.||
AAAAGCUUGAAGACCUUGAAGA
1253
49
|.||.|||||| |.|||...|.||..|.|.|||. ||||||| ||||||.|||.|
CGGAUGUGGAA---GAGAUUUUGUCUAAAGGGCAGCAUUUGUACAAGGAAAAACCAGCC
1255
51
||.||...||||.|||.....|||||||...||| ||.|.|||.|| |.|||.||
CAAACUAGAAAUGCCAUCUUCCUUGAUGUUGGAGGUACCUGCUCUGGCAGAUUUCA
1257
53
|||||..||.|.||.||| |..|..|.|.|||. |.||..|.|||| |||||.|||.|||
AGAACCGGAGGCAACAGUUGAAUGAAAUGUUAAAGGAUUCAACACAAUG-GCUGGAAGCUAAG
1259
55
|||||.|| |||||||.|.|..||
AAAGAGCU-GAUGAAACAAUGGCA
1261
57
||||..|..||||| |||||||
GAAGCCAGUUCUGACCAGUGGAA
1263
59
||.||||| ||.||.||..||||...|||||...|||....| ||| |..|.||.||. |.|||||||.. ||||. ||||..|.|.|.||||||.|.||
AGGCUGAG-GAGGUCAAUACUGAGUGGGAAAAAUUGAACCUGCACU---CCGCUGACUGGCAGAGAAAAAUAGAUGAGACCCUUGAAAGACUCCAGGAACUUCAAGA
1265
60
|||.||.||||.| |..||..||.||..||.|..|||
CAAUGACCUUGCUCGCCAGCUUACCACUUUGGGCAUU
1267
35
|||||..|.|..|| |||||.|||.|||... |||..|||||
GCUACUCAAAAAGAGAUUGAGAAACAGAAGGUGCACCUGAAG
1269
36
||||...|..|||..|.||||| ||||||
GAAACACAUGGAAACUUUUGACCAGAAUG
1271
37
|||.||||||.|.|......|..|.|||...|||||
GAACUGAAUGACAUACGCCCAAAGGUGGACUCUACA
1273
38
|.||.|||||||| |.||.....|.|.||.|...|||...|||||.|... |.||.|.|.||...| ||.|||||||
AAAAUUGCUUGAACCACUGGAGGCUGAAAUUCAGCAGGGGGUGAAUCUGAA-AGAGGAAGACUUCAA--UAAAGAUAUG
1275
39
|||..|||||..||..|| |.|..|....|.|||...|..............||||||| |||| |||.|||..|||.||
AAGACAAUGAGGGUACUGUAAAAGAAUUGUUGCAAAGAGGAGACAACUUACAACAAAGA-AUCA-CAGAUGAGAGAAAGC
1277
41
|||..|||||||...||||. |..||.||.||||.|.| |||.|||
GAUCGGGAAUUGCAGAAGAAGAAAGAGGAGCUGAAUGCAGUGCGUAG
1279
42
|.|.|||||...||||..||| |.||..||||.||
ACACUGUCCGUGAAGAAACGAUGAUGGUGAUGACUG
1281
44
|.||||.|.||| ||||| |.||.||||.|||...||....|.||||.|
ACAAAUUCCUGAGAAUUG----GGAACAUGCUAAAUACAAAUGGUAUCUUAA
1283
46
|||.||||.|||||.||||||
AAAAGAAUAUCUUGUCAGAAU
1285
47
||.|.|||.|..|||....||||||.||.|| ||||.||||....|||..|.|| ||....|||||.|..|.|. |..|||| |||.||.|....|.||.||||
AAUGAAACUGGAGGACCCGUGCUUGUAAGUGCUCCCAUAAGCCCAGAAGAGCAA-GAUAAACUUGAAAAUAAGCUCAAGCAG--ACAAAUCUCCAGUGGAUAAAG
1287
48
|||.|.|||.|||||||
UUGAAAAAAAGCUUGAA
1289
49
|.|||.||||| |||.|.|| ||..|||||.||| ||||||...|.||..|.|..|....|||||
CGGAUGUGGAAGAGAUUUUGUCUAA-AGGGCAGCAUUUGUACAAGGAAAAACCAGCCACUCAGCCAGUGAAG
1291
51
||.||||||...|...||| ||..||.||.|.||||
AGAAAUGCCAUCUUCCUUG-AUGUUGGAGGUACCUG
1293
53
||||||| ||.|.||||.||| |||.|.|..|||....|.|.|.|. ||.||.|.|| |||||| ||.|..|..|||.|||||||||.|||||..||
GAAAGAAUUCAGAAUCAGUGGGAUGAAGUACAAGAACACCUUCAGAACCGGAGGCAACAGUUGAAUGAAAUGUUAAAGGAUUCAACACAAUGGCUGGAAGCUAAGGA
1295
55
||||..|..||||....||..|.|||||...|.|||||.|...|.||.|||.|.||
ACAGAAGCUGAAACAACUGCCAAUGUCCUACAGGAUGCUACCCGUAAGGAAAGGCU
1297
57
|||.||||.|||..|.....||.||..||.. ||.||||.||..|||.||
GGCAGGCACCUAUUGGAGGCGACUUUCCAGC-AGUUCAGAAGCAGAACGA
1299
59
|||.|..||.|||.||||.|| |..|||||.||.| |.||.||.||
UGGCAGAGAAAAAUAGAUGAGACCCUUGAAAGACU--CCAGGAACUU
1301
60
||.||||..|..||||||
AGAAAUUGCGCCUCUGAA
1303
36
||||.|||.||||||
GCAAAAAGAAGACGU
1305
37
||..||||.||.|.|| |||.|||.|.|
UGACCAAGCAGCAAAC-UUGAUGGCAAA
1307
38
|.||..||||||..|.||||...||.|.||| ||.|..|.| ||.|.||.|....|..||..||.|.|||.||.|.||.|.||
GAUAUACAAAAAUUGCUUGAACCACUGGAGGCUGAAAUUCAGCAGGGGGUGAAUCUGAAAGAGGAAGACUUCAAUAAAGAUAUG
1309
39
|.||||..|||||| ||||....|.|||.|...||.||.| |.||.||||||||.|...||.||
GGUACUGUAAAAGA-AUUGUUGCAAAGAGGAGACAACUUACAACAAAGAAUCACAGAUGAGAGAAAG
1311
40
|||||| ||||.|....|....||||..|.|.|.|.|...|||||| |||..||||||
CUCAAAGAAGAAAAAAGGCUCUAGAAAUUUCUCAUCAGUGGUAUCA-----GUACAAGAGGC
1313
41
|.|.||..||||.|.||.| ||||...||||..||||.. |.||||||..| ||...|||...||.|||
GGGAAUUGCAGAAGAAGAAAGAGGAGCUGAAUGCAGUGCGUAGGCAAGCUG-AGGGCUUGUCUGAGGAU
1315
42
|.|.|||||.|....|||..|||||| ||||.|..||
GACUUUGAAGAUCUCUUUAAGCAAGA-GGAGUCUCUG
1317
44
|.|||||.|||. ||.|||..|.| |.|.|||||.|||.||.||||
AUAAAGAUAUUU--AAUCAGUGGCUAACAGAAGCUGAACAGUUUCUCAGA
1319
46
||.|||.||| ||.||.|||||.||...|||
GCAACUAAAAGAAAAGCUUGAGCAAGUCAAG
1321
47
|.|||.||||.||||..|.....|||....|.||||||
UUGAAAAUAAGCUCAAGCAGACAAAUCUCCAGUGGAUA
1323
48
|||||||| ||||.|.|..|||||...| |.|.|||..|.||..|| ||||||||.|...|||
UGAGAAAC--AAGGAGAAAUUGAAGCUCAAAUAAAAGACCUUGGGCAG--CUUGAAAAAAAGCUUG
1325
49
||.||.|||.|.|.|.|.||...|.||.||..|||..|||..|||.|
AAGAGAUUUUGUCUAAAGGGCAGCAUUUGUACAAGGAAAAACCAGCC
1327
51
||||..|.|..|.|..|..|..||||..|.|||.|| |||||..|||.|
AAAAUCACAGAGGGUGAUGGUGGGUGACCUUGAGGA-UAUCAACGAGAU
1329
53
|.||||.||.|||| ||..||||..||||||||
AUACAGUAGAUGCAAUCCAAAAGAAAAUCACAGA
1331
55
|||||||.|...||||||
AAAAGAGCUGAUGAAACA
1333
57
|.||||..|.|||...|.|||||..|....||||.||
UUGGAGGCGACUUUCCAGCAGUUCAGAAGCAGAACGA
1335
59
||||.|. |||...||.|||..|||..||||| |.|..||.|..||.|||.. |||..|||.|| ||||||
UCAAUACUGAGUGGGAAAAAUUGAACCUGCACUCCGCUGACUGGCAGAGAAAAAUAGAUGAGACCCUUGAAA
1337
60
||.|..|||.||||.|...|.|||.|.|.||.|||
UCGAGGAGAAAUUGCGCCUCUGAAAGAGAACGUGA
1339
37
||.|..|.||| ||.|||||| |||.|.||.|||
ACAUACGCCCA-AAGGUGGACUCUACACGUGACCAAG
1341
38
|||||||.| ||.....||.||||||.||.|.|...|.||..||..|||.|.|.|
AGGCUGAAAUUCAGCAGGGGGUGAAUCUGAAAGAGGAAGACUUCAAUAAAGAUAUG
1343
39
|.||||||.|||..| |||||.|......|||..||||
AAGAAUCACAGAUGAGAGAAAGCGAGAGGAAAUAAAGA
1345
40
|||||||| ||..||||||.||
UUGGAUGA--CAUUGAAAAAAAA
1347
41
||||...||||.||||||....|||..||.|.||
GAAUUGCAGAAGAAGAAAGAGGAGCUGAAUGCAG
1349
42
||||...|.||.||.||.|||.|
AGAAGUGGAACAACUUCUCAAUG
1351
44
|||||.|.|...|.||||| |||.|||. |||||.|.|.|.|||..||.|..||..|...||.||||.||
AGUGGCUAACAGAAGCUGA-ACAGUUUC-------UCAGAAAGACACAAAUUCCUGAGAAUUGGGAACAUGCUAAA
1353
46
|||...||||||..|.||||...||||.||
AAAGAGCAGCAACUAAAAGAAAAGCUUGAG
1355
47
||||.||.|.|.||||.|.|.|
AAAAUAAGCUCAAGCAGACAAA
1357
48
|||.||..||||||.||.|.||||
GAAUCAGUUGGAAAUUUAUAACCA
1359
49
|.||.||||..|||||.|
ACAAGGAAAAACCAGCCA
1361
51
|.||..|.||||...||..|||||| ||.||.||
UUGAGGAUAUCAACGAGAUGAUCAU-CAAGCAGA
1363
53
|||||.|..| ||.|||..||||....|||||
CAGAACCGGA-GGCAACAGUUGAAUGAAAUGU
1365
55
|||||..||.|.||..|..|||.|| |.|.||||.||..||.|.|..|||
CAGAAGCUGAAACAACUGCCAAUGU----CCUACAGGAUGCUACCCGUAAGGA
1367
57
||||| |.|||.|..||||..|||.|.|.|.||.|.|| |||.| |||||.|
UGUGG-CUACAGCUGAAAGAUGAUGAAUUAAGCCGGCAGGCACCUAUUGGAGG
1369
59
|.|||..||.|||...||..||..||||| |..|||.|..||| |||.|...||..|| .||||||||||.|.|
AGCAGGCUGAGGAGGUCAAUACUGAGUGGGAAAAAUUGAACCUG-CACUCCGCUGACUG--GCAGAGAAAAAUAGA
1371
60
|||.|||.|||. ||.||||||.|||.||
GAAGACCUGAACACCAGAUGGAAGCUUCUG
1373
38
||.|||.|||..|||.||.||||..||
GAAUUGGAGCAGUUUAACUCAGAUAUA
1375
39
|||.||.||.|||..||||
UUACAGACAAAACAUAAUG
1377
40
|||||||.. |||| ||..|.|.|||||.|.||
AAAUUAGCC-AGCCUACCUGAGCCCAGAGAUGAA
1379
41
|||.||||| |.|||||| |||||.|.|.||.|. ||.|..|.||.|.|...||||
AGUGGAGCC----AACUCAGAUCCAGCUCAGCAAGCGCUG-GCGGGAAAUUGAGAGCAAAUU
1381
42
|||||.||.|...|||.| ||||..||||
AUUAGAAGUGGAACAACUUCUCAAUGCUC
1383
44
|.||.|.|||||..|.|||
AAAGACACAAAUUCCUGAG
1385
46
|||.|..||..||...||..|||. ||||||.|.|||.||||
AGAACAAAAGAAUAUCUUGUCAGA-AUUUCAAAGAGAUUUAA
1387
47
||||.|||||.|.|.|.|...||.||.|||
CAUAAGCCCAGAAGAGCAAGAUAAACUUGA
1389
48
|||.|||...|||..|..||||..|||
UUUCCAGAGCUUUACCUGAGAAACAAG
1391
49
|||||..||..|||..|||
GCAGCAUUUGUACAAGGAA
1393
53
|||||.||||...|||
AAAUCACAGAAACCAA
1395
55
||.|..||.|.|...|.||.||..|.|...||..|||.||.|..|.||..|||||
CAGGAUGCUACCCGUAAGGAAAGGCUCCUAGAAGACUCCAAGGGAGUAAAAGAGC
1397
57
|||.|.|.||||||| ||...|||||.|
GUUCAGAAGCAGAAC-GAUGUACAUAGG
1399
59
|.|||.||||......||..||.||||......||.|||.|||..|||.|||
GAAAAAUUGAACCUGCACUCCGCUGACUGGCAGAGAAAAAUAGAUGAGACCC
1401
60
|||||| |||.| ||...|.||||.|.||.| |.|.||||||...||.|.|.|| |....||..| ||.|||||..|||
ACGUGAGCCACGUCAAUGACCUUGCUCGCCA-----GCUUACCACUUUGGGCAUUCAGCUCUCACCGUAUAACCUCAGCACUC
1403
39
|.||.||.|||||.|..||..| ||||.| ||.|| ||.||..|..|.| ||..||.|.||..|||... ||||||||||
UGUAAAAGAAUUGUUGCAAAGA--GGAGAC--AACUUACAACAAAGAAUCACAGAUGAGAGAAAGCGAGAGGA-AAUAAAGAUA
1406
40
||||| ||||||...||...|..|.|||.|.| |||||.||.||
AGGCUCUAGAAAUUUCUCAUCAGUGGUAUCAGUACAAGAGGCAGGCU
1408
41
||||.|..||||||. |||||..|.|.||.|||||
AAUUUGCUCAGUUUCGAAGACUCAACUUUGCACAAAUU
1410
42
||.||||.|.||.|..||||||
UUAAGCAAGAGGAGUCUCUGAA
1412
44
|||...||.|||||| ||||||.|.|||.|||||.||
GAAGCUGAACAGUUU--CUCAGAAAGACACAAAUUCCU
1414
46
||.||||||.|.| |||||.||..|||.||...|||||
AACAUUGCUAGUAUCCCACUUGAACCUGGAAAAGAGCAG
1416
47
||.||||...|||| ||||||||
AACAAUUAAAUGAA--ACUGGAGG
1418
48
||||||...||| ||||||||.||
UUGAAGACCUUGAAGAGCAGUUAAA
1420
49
|||.||||||.||.|
GAAACUGAAAUAGCA
1422
51
|||.||| |||.||||||
AAAAUCA-CAGAGGGUGA
1424
53
|||||.||||| |.|.||||...|. ||..|||||.||...|||.|.. ||.||||||
UUGAAAGAAUUCAGAAUCAGUGGGAU---GAAGUACAAGAACACCUUCAGA-ACCGGAGGC
1426
55
|.||.|.|||||...|...||..|..||||.||.||||.....||. ||.|||||||
ACUGCAACAGUUCCCCCUGGACCUGGAAAAGUUUCUUGCCUGGCUUACAGAAGCUGAAA
1428
57
|||.|||.|..|.||.|..|.||||||||.||.||.||.|
GAACUUCUGGUGUGGCUACAGCUGAAAGAUGAUGAAUUAA
1430
59
|||.|||.||| ||||..| ||||.|.||.||
AGGAGGUCAAUACUGAGUG-GGAAAAAUUGAA
1432
60
||||||||||.|.|
UCUGAAAGAGAACG
1434
40
||||.||||..||.|||||
GAGAUGAAAGGAAAAUAAA
1436
41
||.||||.|||||.|.|.|.| || ||.|.|.| |.|.||||..|||||.||||
AGUGGAGCCAACUCAGAUCCAGCUCAGCAAGCGCUG--GCGGGAAAUUGAGAGCAAAU
1438
44
|.||.|||||||||....|.|||..|.|| ||||
AUGAUAUAAAGAUAUUUAAUCAGUGGCUAACAGA
1440
46
|||.|..||.|.||.|.||...|..||| |||||..|||.||.|.|||
AACCUGGAAAAGAGCAGCAACUAAAAGA-AAAGCUUGAGCAAGUCAAG
1442
47
||||.|.|.|.|||.||||.....||||.||||....|.|.|....|||.. ||.||.|.|.....|||.|..|| |||||..|.||| |..||||....||||||||.|
AGGGAAUUCUCAAACAAUUAAAUGAAACUGGAGGACCCGUGCUUGUAAGUGCUCCCAUAAGCCCAGAAGAGCAAG---AUAAACUUGAAA-AUAAGCUCAAGCAGACAAAUC
1444
48
||||||....|||.||.|.|||.|.|||.|..||
GAGAAACAAGGAGAAAUUGAAGCUCAAAUAAAAG
1446
49
||.||.|||.||..|..||||.|.||......|||||..||.||.||
UGGAAGAGAUUUUGUCUAAAGGGCAGCAUUUGUACAAGGAAAAACCA
1448
51
|.|.|||||||||.| |.||..|.|.|.|....|.|.||||
AUAAAAUCACAGAGG-GUGAUGGUGGGUGACCUUGAGGAUA
1450
53
|||..||| ||.|| ||.|...|||.|||||....|.|| ||..||||.|||| ||..|..|||...|.|..|||...|.|.|..|...||||. |||.||..||.| |||||.|.|||
AAUUCAGAAUCAGUG- GGAUGAAGUACAAGAACACCUUCAGAACCGGAGGCAACAGUUGAAUGAAAUGUUAAAGGAUUCAACACAAUGGCUGGAAG CUAAGGAAGAAGCUGAGCAGGUCUUA
1452
55
||||||....||||. ||||||||..||.||.||
GAAGACUCCAAGGGA---GUAAAAGAGCUGAUGAAA
1454
57
|.|||||...|||.||.|||
GCUACAGCUGAAAGAUGAUG
1456
59
||.||||..|.||||||||
CAGAGAAAAAUAGAUGAGA
1458
60
|||.|||.|.||.|||
UGAAAGAGAACGUGAG
1460
41
|.|||...||..|||.|...||.|||||.|..|||||.||
AGCUCAGCAAGCGCUGGCGGGAAAUUGAGAGCAAAUUUGC
1462
42
|.|.|.|.||.|.|||||||||.||
AGACAUGCCUUUGGAAAUUUCUUAU
1464
44
|.|.|.||.|.||.|.|.|||||||| ||.|||.|
AUGAUAUAAAGAUAUUUAAUCAGUGGCUAACAGAA
1466
46
CCUACCUGAGCCCAGAGAUGAAAGGAAAAUAAA
||||..|..|...|||.|||||.||. |.|.|.||..|.||.|| ||.||.|||.||..||.|.||...|.||.||||
AUGAAUUUGUUUUAUGGUUGGAGGAA--- GCAGAUAACAUUGCUAGUAUCCCACUUGAACCUGGAAAAGAGCAGCAACUAAA
1468
47
|||...|||||.|.||||.||.| |....||..||||||
AAACUUGAAAAUAAGCUCAAGCAGACAAAUCUCCAGUGG
1470
48
|||.|||||.| |||.|||||.|..|.|||
AAAAGCUUGAA-GACCUUGAAGAGCAGUUA
1472
49
|||.|.||..|..|.||||||. ||.|||| ||.||.|
UUGUACAAGGAAAAACCAGCCA--CUCAGCC-AGUGAAG
1474
51
|||.||||.||||
UGGGUGACCUUGA
1476
53
||||....||||||||.||.| ||||||||. ||...|.|..|.|....|.||...|||.||||.||...|||
AAUUCAGAAUCAGUGGGAUGAAGUACAAGAA-CACCUUCAGAACCGGAGGCAACAGUUGAAUGAAAUGUUAAA
1478
55
|.||.|.|||.|||. ||.||||..|||...||||
UAGAAGACUCCAAGGGAGUAAAAGAGCUGAUGAAA
1480
57
||..|.||||.|.||||||.|.|
AGCCAGUUCUGACCAGUGGAAGC
1482
59
|.||||...||||...||..|...|..|.|||||.|..|.|..||...|||..|.|.|||....|.||.|....|.|||||.|......|||.|.| ||||.
ACGAAAGCAGGCUGAGGAGGUCAAUACUGAGUGGGAAAAAUUGAACCUGCACUCCGCUGACUGGCAGAGAAAAAUAGAUG AGACCCUUGAAAGACU--CCAGGAA//
||.|..|||.|||..|||||...|.|..|.|||
//CUUCAAGAGGCCACGGAUGAGCUGGACCUCAAG
1484
60
|||||||.|.|.||. |.|..||.||..|||.||.|...||||.||
AGAAAUUGCGCCUCU---GAAAGAGAACGUGAGCCACGUCAAUGACCU
1486
42
|||.|||||||| |||||
AAGCAAGAGGAGUCUCUGAA
1488
44
||..||..|||||||. |||||.|||.||
AAGCUGAACAGUUUCUCAGAAAGACACAAAUU
1490
46
|||.|.|| ||.|.||.||.|.|..||.|..|||||
GAAAAGAGCAGCAACUAAAAGAAAAGCUUGAGCAAG
1492
47
|||||||....||.||..||..|.|.|||.|.| |.||||.|
GGGAAUUCUCAAACAAUUAAAUGAAACUGGAGGACCCGUGCUU
1494
48
|.|||.|||||..|.||||.||
ACAAGGAGAAAUUGAAGCUCAA
1496
49
|||.||.||||.|.|||.||...|.||.|.|
CAGUUCAAGCUAAACAACCGGAUGUGGAAGA
1498
53
|||.||.| ||| |||.|||| |||||| ||||..|.||||..|| |.|||. |||||..|.| |||.|| ||.|.|.||. ||||.||..|||..||.|
AAUGGCUGGAAGCUAAGGAAGA--AGCUGA-- GCAGGUCUUAGGACAGGCCAGAGCCAAGCUUGAGUCAUGGAAGGAGGGUCCCUAUA-CAGUAGAUGCAAUCCAAA
1500
55
||.||||||..||||.|
UUACAGAAGCUGAAACA
1502
57
||.||..|.||...||..||||||.|| |||||..||...|.|||||
UCUGGUGUGGCUACAGCUGAAAGAUGA--UGAAUUAAGCCGGCAGGCA
1504
59
|.||||.|.||..||...|..||.||||||.|......||...|.||||||||||
CAGGAACUUCAAGAGGCCACGGAUGAGCUGGACCUCAAGCUGCGCCAAGCUGAGG
1506
60
||||||..||| ||.|..|||| ||.|||
GAGCCACGUCAAUGACCUUGCUC-GCCAGC
1508
44
|||..|||||..|||||..||||||
CUAACAGAAGCUGAACAGUUUCUCA
1510
46
|.|.||..||..|.||.|||..|..|....|.|||.|.|.|||||
UAACAUUGCUAGUAUCCCACUUGAACCUGGAAAAGAGCAGCAACU
1512
47
||.||.|||||| |..|||....||..|||||...|||
UCCCAUAAGCCC-AGAAGAGCAAGAUAAACUUGAAAAU
1514
48
|.|.|||.|||||||.|..||.||.||
UGAAGACCUUGAAGAGCAGUUAAAUCA
1516
49
|||.|.|.|.||||||
CACUCAGCCAGUGAAG
1518
51
|.|.|.|.||||||||. |||| |||.||.||
ACAGAGGGUGAUGGUGGGUGACCUUGAGGAUAU
1520
53
||||.||.|.|..|...||.||.||.|.|||..| ||.|.||||.|||
CACAAUGGCUGGAAGCUAAGGAAGAAGCUGAGCAGGUCUUAGGACAGGCC
1522
55
||||| ||.|||||||.| ||||| ||||
AGAAGCUGAAACAACUGC-CAAUG-UCCU
1524
57
|.||.||||..||||.||.|..||.|
GGAACUUCUGGUGUGGCUACAGCUGA
1526
59
||||.|.|.|||.||..|||||
ACUGGCAGAGAAAAAUAGAUGA
1528
60
||||...||.|.||.| |||||..|.||..|.|.. |||.|..||||||
AGAACGUGAGCCACGU--CAAUGACCUUGCUCGCCA-GCUUACCACUUUG
1530
44
|.||||||.|.|.||| ||||..|||
AUUGGGAACAUGCUAAAUACAAAUGGUA
1532
46
|||.|.||||||| .|.||..|.||.||.||.|.||
GAAAAGAGCAGCA--ACUAAAAGAAAAGCUUGAGCAA
1534
47
||||||..||..||.| |||||||.|
AAGAUAAACUUGAAAAUAAGCUCAAG
1536
48
|.|.|||||.|....||.| ||.|.|||||....||||.||....||.|.||..|||.|
GUUAUCUCCUAUUAGGAAU--CAGUUGGAAAUUUAUAACCAACCAAACCAAGAAGGACCA
1538
49
||||...|..|..||||||||||...||.||
AAAGGGCAGCAUUUGUACAAGGAAAAACCAG
1540
51
|.|||.|.|.|||| |.|| ||||.||.....|.|.||.|| ||.||.|.|||||||
UGGCAGAUUUCAACCGGGCU-UGGACAGAACUUACCGACUGG-----CUUUCUCUGCUUGAU
1542
53
||.|||.|...||...||.||.|..|.||..||||||..|||||
CACCUUCAGAACCGGAGGCAACAGUUGAAUGAAAUGUUAAAGGA
1544
55
|||.||.|.|..|||||||.|
UGCUACCCGUAAGGAAAGGCU
1546
57
||||..|||||||
GGUGUGGCUACAG
1548
59
|.|||||||||.|.|||
AGUGGGAAAAAUUGAAC
1550
60
||.|||||....||||. |.|.| |||||||
CUCUGGAAGACCUGAACACCAGAUGGAAGCU
1552
45
|.||||.|.|| |..|||.|| ||| |||||.|. |||||.|||.|.||
UGAAUCUGCGG-UGGCAGGAGGUCUGCAAACAGCUG-UCAGACAGAAAAAA
1554
54
|||||||..|||.|.|..||
CAGUGGCAGACAAAUGUAGA
1556
56
|.|.||.||.|||.||||||||
AAACAGCCAAAAAAUCCUGAGA
1558
46
||||||. .||||||...||.|||||
GCAACUA--AAAGAAAAGCUUGAGCAA
1560
47
|.|||.||.....||.|||.|..|||.||||.||||||.|.|
CUGCGCCAGGGAAUUCUCAAACAAUUAAAUGAAACUGGAGGA
1562
48
|||||.|||.||||
UUGGGCAGCUUGAA
1564
49
|.|.|.|||.|||. |||| |||||||
AAAGGGCAGCAUUUGUACAAGGAAAAA
1566
51
|.||.|||..||||. |.|.||||||
CUGCCAUCUCCAAACUAGAAAUGCCA
1568
53
||||||| .|.|.|||||.|..||| |||||..|..||.|.|| ||.||| |||.|.|.|..||| |||||.||
UCAGAAC--CGGAGGCAACAGUUGAAUGAAAUGUUAAAGGAUUC----AACACA-AUGGCUGGAAGCUA-AGGAAGAA
1570
55
|||||..|||||.|.|.||||||||
AAACAACUGCCAAUGUCCUACAGGA
1572
57
|||.|||||||.|. |||| |.||||.|| .|||||||
AUUUGGAAGCCAGU-UCUGACCAGUGGAAG--CGUCUGCA
1574
59
|||||..|.||||.|.|.||||.||||
CUGCACUCCGCUGACUGGCAGAGAAAA
1576
60
|||.||.|.||||||..||. ||||.|||
CCUGAACACCAGAUGGAAGC-UUCUGCAG
1578
54
||..|||..|..||||||..||.....|||.|||.||
CAGAUGAUACCAGAAAAGUCCACAUGAUAACAGAGAA
1580
56
||||||||..|.|.||||.|..|||
AACCUGGAUGAAAACAGCCAAAAAA
1582
48
|.||||.|..|...|.||||||||.|||||
AAAAGACCUUGGGCAGCUUGAAAAAAAGCU
1584
49
|||||..||.|..||.||||||..|..||.|....|||||
AAACUGAAAUAGCAGUUCAAGCUAAACAACCGGAUGUGGA
1586
51
|.||.|||..||..||.| |||||||||
AUAUCAACGAGAUGAUCA--UCAAGCAGA
1588
53
||||.||.|||||||
AACAGUUGAAUGAAA
1590
55
||.||.||||.||||||..||.||.||
UGCAACAGUUCCCCCUGGACCUGGAAA
1592
57
|.||..|||||||.|| |||.|||. |..||||.|.||||
UGACCAGUGGAAGCGUCUGCACCUUUCUCUGCAGGAACUUCU
1594
59
||||| |||| ||.|.|||..||||| |.|.|.|..||....|.|..|||.|.||.||..|. ||||.|||. ||||...||...||..||...|.| ||.|.||.||....|||||
UACUGAGUGGGAAAAAUUGAACCUGC-------ACUCCGCUGACUGGCAGAGAAAAAUAGAUGAGACC--CUUGAAAGA-CUCCAGGAACUUCAAGAGGCCACGGAUGAGCUGGACCUCAAGCUGCGC//
|||||.||
//CAAGCUGA
1596
60
|.|.|||..|.||||.|||..|..||.|||.. |.|..||..|.|||||
UCGAGGAGAAAUUGCGCCUCUGAAAGAGAACG-UGAGCCACGUCAAUGA
1598
49
|||||.|.||.||||. ||||..|||
GAAAUAGCAGUUCAAGCUAAACAACC
1600
51
||..|||||.||..||....|||||.|.|
CUCUGGCAGAUUUCAACCGGGCUUGGACA
1602
53
|.||||.||. ||| |||.|..|||..||..|..|..|||...||||.| |||||..|||..||.|||
UCAGAAUCAGUGGGAUGAAGUACAAGAACACCUUCAGAACCGGAGGCAACAGUUGAAUGAAAUGUUAAAG
1604
55
|.|.||.|||||....||||.....||| |.||.||.|.||.||.||||...|||
GGACCUGGAAAAGUUUCUUGCCUGGCUU--ACAGAAGCUGAAACAACUGCCAAUGU
1606
57
|.||||.|..|...||.||||..||.|.|||||..||.|
UGGAAGCCAGUUCUGACCAGUGGAAGCGUCUGCACCUUU
1608
59
|||.||||||||.||..|.|.......|| |||||. ||||||.||.| ||...|||||||
GGAAAAAUUGAACCUGCACUCCGCUGACU--GGCAGA--GAAAAAUAGAU-GAGACCCUUGAA
1610
60
|.|||||||||||...|||..| ||.|||.|.||.||..|.|.||
CGAGGAGAAAUUGCGCCUCUGA-AAGAGAACGUGAGCCACGUCAA
1612
51
|||||||...|||| |||..||
CAACCGGGCUUGGACAGAACUU
1614
53
||||.||..||..||. ||..|.|||||||.|
UGAAGUACAAGAACAC-CUUCAGAACCGGAGG
1616
55
||||||.|.|.|..|.|.|||......|..|.|||..|||||.||.|| |||.||
GAAACUCAUAGAUUACUGCAACAGUUCCCCCUGGACCUGGAAAAGUUUCUUGCCU
1618
57
||||||||| |||..|||.|
AGCAGUUCAGAAGCAGAACGA
1620
59
|||||||...| ||.|.||.|||. |||.|||..||.|
UCAAGCUGCGCCAAGCUGAGGUGAUCAAGGGAUCCUGGC
1622
60
|||.|||.||...||....||..|.||.||||||
GCACUUCGAGGAGAAAUUGCGCCUCUGAAAGAGA
1624
51
||||||.||.|||..||. |.||| |.||.||...|||.|.. |..|||| |||.|||.....||||||
AAGUUAUAAAAUCACAGA---GGGUG-AUGGUGGGUGACCUUGAGGAUAUCAACGAGAUGAUCAUCAAGCAG
1626
53
||..||||..|.||.. |.|||||.|.|..|||.|.......|||||..|.|.||||..|.||..|..|||..| |.|...|||.|.|.|||.||.|
UUGAAAGAAUUCAGAA-UCAGUGGGAUGAAGUACAAGAACACCUUCAGAACCGGAGGCAACAGUUGAAUGAAAU-GUUAAAGGAUUCAACACAAUGG
1628
55
|||.|||.| ||||.||||
AGGGAGUAA-AAGAGCUGA
1630
57
||.|||||.| ||||||||....|..|||.||..||.||.||.||
AGUUCUGACC----AGUGGAAGCGUCUGCACCUUUCUCUGCAGGAACU
1632
59
||||||||||...|.||...|||.| ||||| ||||.|
ACUUCAAGAGGCCACGGAUGAGCUG---GACCUCAAGCUGC
1634
60
||||||.||||.....||.||||||.|.||
UGACCUUGCUCGCCAGCUUACCACUUUGGG
1636
52
||.|.||..|.|.||.||.|||.||..||
AUUUGAAAAACAAGACCAGCAAUCAAGAG
1638
58
|||||.|||.|||..|||
ACUAAAGAACCUGUAAUC
1640
53
|||.|||||.|||..|.|..||.....||.|..||.|||| |||.|.|.|..|||.||...|....|||..|.|.||||..||
UUGAAAGAAUUCAGAAUCAGUGGGAUGAAGUACAAGAACACCUUCAGAACCGGAGGCAACAGUUGAAUGAAAUGUUAAAGGAUUCAA
1642
55
||||||||| |||||
UUGGAAGAAACUCAU
1644
57
||||||.|..|||.|| |||||.||
CCAGCAGUUCAGAAGC-AGAACGAU
1646
59
||||||.||. ||||.|| |||||||||.|
UUGAAAGACU---CCAGGAACUUCAAGAGGCCA
1648
60
|||||||.||.|..|||
UGGAAGACCUGAACACC
1650
58
|.||||||. |||.|||..|.|.||||
CAGAGCAGC-CUUUGGAAGGACUAGAG
1652
55
|||||||..||.| .|.|.|.||.||| |||.||||.|.||.|
CUGAAACAACUGC--CAAUGUCCUACAGGAUGCUACCCGUAAGG
1654
57
|||..|||||...|||||.||
CAGAAGCAGAACGAUGUACAU
1656
59
||||||. |.|||||||.|||..||||||.|.|...|||......||.|.||||||
CUCCGCUGACUGGCAGAGAAAAAUAGAUGAGACCCUUGAAAGACUCCAGGAACUUC
1658
60
|.||||..||..|||.||....||....|.||| ||||.|.||.|...|.|||
UGCGCCUCUGAAAGAGAACGUGAGCCACGUCAA-UGACCUUGCUCGCCAGCUU
1660
56
||||||.|.|||..||...||..|...|.|||||..|.|.|..|..|||..|| |||.||||..|||.|.|
GAAGCUCACACAGAUGUUUAUCACAACCUGGAUGAAAACAGCCAAAAAAUCCUGAGAUCCCUGGAAGGUUCCGAUG
1662
57
|||||..|||.||..|||..|.. |.||.|||..||.||
ACAGCUGAAAGAUGAUGAAUUAAGCCGGCAGGCACCUAU
1664
59
||..||||||||.|| |||.|.|.||..|..||.| |..|.|||||.|.|....|||.|.|||.|..|...|..||.|...|.|||||
GGAAAAAUUGAACCUGCACUCCGCUGACUGGCAGAGAAAAAUAGAUGAGACCCUUGAAAGACUCCAGGAACUUCAAGAGGCCACGGAUGA
1666
59
|.|||..||.|..|.|||.||||..||.||||..|..|||
AUAGAUGAGACCCUUGAAAGACUCCAGGAACUUCAAGAGG
1668
60
||||.||..|.|||||
CUUCGAGGAGAAAUUG
1670
60
|..|.|||.|||||||...|||..|.|||
CUUCGAGGAGAAAUUGCGCCUCUGAAAGA
1743
18
|||..|||.|||||
CAGACUUAAAAGAA
1767
18
||||..|||..|..|||||
AUUUGCAAUCUUUCGGAAG
1769
30
||.||||.||||..| |.|||||||
CAGUUGGCAGCUUAUAUUGCAGACAAG
1771
30
|||||.||||. ||.|||....|.|.|||.|..||
GGAGACUGAAA-AAUCCUUACACUUAAUCCAGGAG
1773
55
|.||..||..||||||||
AGGCUGCUUUGGAAGAAA
1775
55
|||..||..||||..|||.||.|..||
AGCGAGAGGCUGCUUUGGAAGAAACUC
1777
Перечень предпочтительных последовательностей оснований олигонуклеотидов по изобретению. Олигонуклеотиды, содержащие эти последовательности оснований, способны связываться с высоко сходными областями (участки последовательности с >50% идентичностью) в двух различных экзонах DMD (как проиллюстрировано в таблице 2), и они способны индуцировать пропускание этих двух экзонов и любых экзонов между ними с образованием транскрипта DMD в рамке считывания.
Перечень наиболее предпочтительных и/или дополнительных комбинаций экзонов, наиболее предпочтительных и/или дополнительных областей идентичности экзонов, и наиболее предпочтительных и/или дополнительных олигонуклеотидов с их последовательностями оснований, химическими модификациями и идентификационными номерами последовательностей, где *= LNA, C=5-метилцитозин, U=5-метилурацил, D=2,6-диаминопурин, X=C или 5-метилцитозин, Y=U или 5-метилурацил, Z=A или 2,6-диаминопурин, и < > все 2'-фторнуклеотиды (как в SEQ ID NO: 1844).










Примеры 1-5
Материалы и способы
Конструирование олигонуклеотидов в первую очередь было основано на обратной комплементарности с конкретными высоко сходными участками последовательности в двух различных экзонах DMD, как идентифицировано в EMBOSS Matcher и как описано в таблице 2. Следующими учитываемыми параметрами последовательности были присутствие частично открытых/закрытых структур РНК в указанных участках последовательности (как предсказано с помощью RNA structure версии 4.5 или РНК mfold версии 3.5 (Zuker, M.) и/или присутствие предполагаемых участков связывания SR-белка в указанных участках последовательности (как предсказано с помощью программного обеспечения ESE-finder (Cartegni L, et al. 2002 и Cartegni L, et al., 2003). Все AON были синтезированы в Prosensa Therapeutics B.V. (Leiden, Netherlands) или были получены из коммерческого источника (ChemGenes, США), и они содержат 2'-O-метил-РНК и полноразмерные фосфоротиоатные (PS) основные цепи. Все олигонуклеотиды представляли собой 2'-O-метилфосфоротиоатную РНК, и их синтезировали в масштабе 10 мкмоль с использованием устройства для синтеза OP-10 (GE/ÄKTA Oligopilot), с помощью стандартных протоколов для фосфорамидитов. Олигонуклеотиды расщепляли и из них удаляли защитные группы двухстадийным способом (обработка DIEA, а затем концентрированным NH4OH), очищали с помощь ВЭЖХ и растворяли в воде, и добавляли избыток NaCl для обмена ионами. После упаривания соединения перерастворяли в воде, обессоливали с помощью FPLC и лиофилизировали. Масс-спектрометрия подтвердила идентичность всех соединений, и чистота (определенная с помощью UPLC) была найдена приемлемой для всех соединений (>80%). Для экспериментов in vitro, описанных в настоящем описании, приготавливали 50 мкМ рабочие растворы AON в фосфатном буфере (pH 7,0).
Культивирование тканей, трансфекция и анализ способом ОТ-ПЦР
Дифференцированные здоровые контрольные мышечные клетки человека (мышечные трубочки) трансфицировали в 6-луночных планшетах при стандартной концентрации AON 400 нМ в соответствии со стандартными рабочими методиками, не относящимися к GLP. Для трансфекции использовали полиэтиленимин (ExGen500; Fermentas Netherlands) (2 мкл на мкг AON, в 0,15 M NaCl). Упомянутые выше методики трансфекции были адаптированы из ранее описанных материалов и способов (Aartsma-Rus A., et al., 2003). Через 24 часа после трансфекции РНК выделяли и анализировали способом ОТ-ПЦР. В кратком изложении, для получения кДНК использовали случайную смесь гексамеров (1,6 мкг/мкл; Roche Netherlands) в реакции с обратной транскриптазой (RT) на 500-1000 нг исходной РНК. Затем проводили анализ ПЦР на 3 мкл кДНК для каждого образца, и он включал первую и гнездовую ПЦР с использованием праймеров, специфичных к гену DMD (см. таблицы 4 и 5). Выделение РНК и анализ ОТ-ПЦР проводили в соответствии со стандартными рабочими методиками, не относящимися к GLP, адаптированными из ранее описанных материалов и способов (Aartsma-Rus A., et al., 2002; и Aartsma-Rus A., et al., 2003). Продукты ОТ-ПЦР анализировали гель-электрофорезом (2% агарозные гели). Полученные фрагменты ОТ-ПЦР количественно определяли с помощью анализа DNA Lab-on-a-Chip analysis (Agilent Technologies USA; DNA 7500 LabChips). Данные обрабатывали с помощью программного обеспечения "Agilent 2100 Bioanalyzer" и Excel 2007. Оценивали соотношение меньших продуктов транскрипции (содержащих ожидаемое пропускание множества экзонов) и общего количества продуктов транскрипции (что соответствует эффективности пропускания в процентах), и прямо сравнивали с количеством продуктов транскрипции в нетрансфицированных клетках. Фрагменты ПЦР также выделяли из агарозных гелей (набор для экстракции из геля QIAquick gel extraction kit, Qiagen Netherlands) для подтверждения последовательности (секвенирование по методу Сенгера, BaseClear, Нидерланды).
Наборы праймеров для ПЦР, использованные для обнаружения пропускания экзонов-мишеней
Последовательности праймеров
Результаты
Пример 1. Нацеливание на участок последовательности с высоким сходством в экзонах 10 и 18 посредством AON в различных положениях
Исходя из высоко сходного участка последовательностей (63%) в экзонах 10 (SEQ ID NO: 109) и 18 (SEQ ID NO: 110) конструировали серию AON, распределенных по указанному участку последовательности, на 100% обратно комплементарные либо экзону 10 (PS814; SEQ ID NO: 1675, PS815; SEQ ID NO: 1677, PS816; SEQ ID NO: 1679) или либо экзону 18 (PS811; SEQ ID NO: 1673). После трансфекции в контрольных культурах мышечных трубочек здорового человека анализ ОТ-ПЦР продемонстрировал, что все четыре AON были способны индуцировать пропускание экзонов с 10 по 18 (что подтверждено с помощью анализа последовательности) (фиг.1). PS811 и PS816 обладают наиболее высокими процентами обратной комплементарности с обоими экзонами (фиг. 1B), и они были наиболее эффективными с эффективностью пропускания экзонов с 10 по 18, составляющей 70% и 66%, соответственно (фиг.1C). PS814 был наименее эффективным, что может быть связано с его расположением и/или более короткой длиной (21 против 25 нуклеотидов) и, таким образом, более низкой аффинностью или стабильностью. В необработанных клетках не наблюдали пропускания экзонов с 10 по 18 (NT). Эти результаты были высоко воспроизводимыми (пропускание экзонов с 10 по 18 посредством PS816 при 20/20 различных трансфекциях) и демонстрируют, что пропускание мультиэкзонного участка с 10 по 18 экзоны является осуществимым с использованием одного AON, который способен связываться как с экзоном 10, так и с экзоном 18, в области с высоким сходством последовательности (63%), и который способен индуцировать пропускание этих экзонов и всех экзонов между ними. Хотя в этой области транскрипта могут быть получены дополнительные фрагменты с пропусканием множества экзонов, полученный транскрипт, в котором экзон 9 был прямо сплайсирован с экзоном 19 (транскрипт в рамке считывания), был наиболее распространенным во всех экспериментах.
Пример 2. Нацеливание на участок последовательности с высоким сходством в экзоне 10 и 18 посредством AON различной длины
Учитывая высоко сходные участки последовательности в экзонах 10 (SEQ ID NO: 109) и 18 (SEQ ID NO: 110), авторы настоящего изобретения оценивали эффект AON различной длины для идентификации наиболее эффективной минимальный длины. После трансфекции PS816 (25-мер; SEQ ID NO: 1679), PS1059 (21-мер; SEQ ID NO: 1684) или PS1050 (15-мер; SEQ ID NO: 1682) в контрольных мышечных клетках здорового человека (т.е. дифференцированных мышечных трубочках), анализ ОТ-ПЦР продемонстрировал, что все три AON были способны индуцировать пропускание экзонов с 10 по 18 (что подтверждено с помощью анализа последовательности) (фиг.2A, B). PS816 и PS1059 были наиболее эффективными (68% и 79%, соответственно). Наиболее короткий PS1050 был менее эффективным (25%). В необработанных клетках (NT) не наблюдали пропускания экзонов с 10 по 18. Эти результаты указывают на то, что пропускание мультиэкзонного участка с 10 по 18 экзоны является осуществимым с использованием AON из 15, 21 и 25 нуклеотидов. Также ожидается, что будут действовать более длинные AON. 21-мерный PS1059 был наиболее эффективным наиболее коротким кандидатом. 15-мер PS1050 был наименее эффективным, что может быть связано с его уменьшенной обратной комплементарностью экзону 18 (47%; фиг.2B) и/или уменьшенной аффинностью связывания с РНК-мишенью или стабильностью. Для повышения Tm и, таким образом, аффинности связывания и стабильности дуплекса, 15-меров в целях повышения их биоактивности могут требоваться модификации оснований. Наиболее многочисленным полученным продуктом-транскриптом в этом эксперименте вновь был продукт, в котором экзон 9 был прямо сплайсирован с экзоном 19 (транскрипт в рамке считывания).
Пример 3. Нацеливание на участок последовательности с высоким сходством в экзоне 10 и 18 посредством AON с модифицированной химической структурой оснований
Конкретные характеристики выбранной химической структуры AON, по меньшей мере частично, влияет на доставку AON к транскрипту-мишени: путь введения, биостабильность, биораспределение, внутритканевое распределение и клеточный захват и транспорт. Кроме того, считается, что дальнейшая оптимизация химической структуры олигонуклеотида повысит аффинность связывания и стабильность, повысит активность, улучшит безопасность и/или снизит затраты вследствие уменьшения длины или усовершенствования методик синтеза и/или очистки. В высоко сходном участке последовательностей в экзонах 10 (SEQ ID NO: 109) и 18 (SEQ ID NO: 110) авторы настоящего изобретения оценивали эффект AON на основе 2'-O-метил-РНК с различными модифицированными основаниями (такими как 5-замещенные пиримидины и 2,6-диаминопурины). После трансфекции PS816 (AON с регулярной 2'-O-метил РНК; SEQ ID NO: 1679), PS1168 (PS816, но в котором все As заменены на 2,6-диаминопурин; SEQ ID NO: 1681), PS1059 (AON с регулярной 2'-O-метилфосфоротиоатной РНК; SEQ ID NO: 1684), PS1138 (PS1059, но в котором все Cs заменены на 5-метилцитозин; SEQ ID NO: 1685), или PS1170 (PS1059, но в котором все As заменены на 2,6-диаминопурин; SEQ ID NO: 1686) (фиг.3B) в контрольных мышечных клетках здорового человека (т.е. дифференцированные мышечные трубочки). Анализ ОТ-ПЦР продемонстрировал, что все пять AON были способны индуцировать пропускание экзонов с 10 по 18 (что подтверждено с помощью анализа последовательности) (фиг.3). PS816 был наиболее эффективным (88%). Хотя модификации оснований в этих конкретных последовательностях не повышали биоактивность еще более, они могли иметь более положительный эффект на биораспределение, стабильность и/или безопасность, так что менее эффективные соединения все еще являются предпочтительными для клинической разработки. Эти результаты указывают на то, что пропускание мультиэкзонного участка с 10 по 18 экзоны является осуществимым с использованием AON с модифицированными основаниями. Наиболее многочисленным полученным транскриптом-продуктом в этом эксперименте вновь был транскрипт, в котором экзон 9 был прямо сплайсирован с экзоном 19 (транскрипт в рамке считывания).
Пример 4. PS816 индуцирует пропускание других мультиэкзонных участков в рамке считывания
Высоко сходный участок последовательности в экзонах 10 (SEQ ID NO: 109) и 18 (SEQ ID NO: 110) также (частично) присутствует в экзонах 30 (SEQ ID NO: 128), 31 (SEQ ID NO: 130), 32 (SEQ ID NO: 132), 42 (SEQ ID NO: 152), 47 (SEQ ID NO: 158), 48 (SEQ ID NO: 160), 57(SEQ ID NO: 170) и 60 (SEQ ID NO: 174) (фиг.4A, B, таблица 2). Таким образом, авторы настоящего изобретения сфокусировались на выявлении пропускания мультиэкзонных участков после трансфекции 400 нМ PS816 (SEQ ID NO: 1679). Авторы настоящего изобретения использовали различные наборы праймеров (таблица 4 и 5) для этой цели. Действительно, с помощью анализа ОТ-ПЦР авторы настоящего изобретения выявили пропускание в рамке считывания экзонов с 10 по 30, экзонов с 10 по 42, экзонов с 10 по 47, экзонов с 10 по 57 и/или экзонов с 10 по 60 в множественных экспериментах (подтверждено с помощью анализа последовательности), которое не наблюдалось в необработанных клетках. В качестве примера, на фиг.4C представлено индуцированное PS816 пропускание экзонов с 10 по 18, экзонов с 10 по 30 и экзонов с 10 по 47. Несмотря на дополнительные случаи пропускания множества экзонов (как в рамке считывания, так и не в рамке считывания), полученный транскрипт, в котором экзон 9 был прямо сплайсирован с экзоном 19 (транскрипт в рамке считывания), был наиболее воспроизводимым и оказался наиболее многочисленным во всех экспериментах. Эти результаты подтверждают, что в гене DMD может быть индуцировано пропускание множества экзонов с использованием одного AON, который способен связываться с участком последовательности, который высоко сходен между двумя различными экзонами и способен индуцировать пропускание этих экзонов и всех экзонов между ними с образованием транскрипта DMD в рамке считывания.
Пример 5. Нацеливание на участок последовательности с высоким сходством в экзоне 45 и 55 с помощью AON с модифицированной химической структурой оснований
Исходя из высоко сходного (80%) участка последовательности в экзонах 45 (SEQ ID NO: 1571) и 55 (SEQ ID NO: 1572) была сконструирована серия AON, распределенных по всему указанному участку последовательности, либо на 100% обратно комплементарных экзону 45 (PS1185; SEQ ID NO: 1706, PS1186; SEQ ID NO: 1707), либо на 96% обратно комплементарных экзону 55 (с одним несоответствием оснований) (PS1188; SEQ ID NO: 1713) (фиг.5A). Во всех трех AON As были заменены на 2,6-диаминопурин. После трансфекции в контрольных культурах мышечных трубочек здорового человека анализ ОТ-ПЦР продемонстрировал, что PS1185, PS1186 и PS1188 были способны индуцировать пропускание экзонов с 45 по 55 (что подтверждено с помощью анализа последовательности) (фиг.5B). В необработанных клетках (NT) не наблюдали пропускания экзонов с 45 по 55. Эти результаты демонстрируют, что пропускание другого мультиэкзонного участка с 45 по 55 экзоны является осуществимым с использованием одного AON, который способен связываться как с экзоном 45, так и с экзоном 55 в области с высоким сходством последовательности (80%), и который способен индуцировать пропускание этих экзонов и всех экзонов между ними. Полученный транскрипт, в котором экзон 44 был прямо сплайсирован с экзоном 56, находится в рамке считывания, и он был выявлен во множестве экспериментов. Что касается экспериментов с пропусканием экзонов с 10 по 18 (пример 3), авторы настоящего изобретения вновь продемонстрировали, что можно эффективно использовать AON с модифицированными основаниями. Более того, результаты, полученные с помощью PS1188, указывают на то, что AON не должен быть на 100% обратно комплементарным одному из экзонов-мишеней, но также может быть сконструирован в качестве гибридных структур, в которых отсутствует 100% обратная комплементарность с каким-либо экзоном-мишенью.
Описание чертежей
Фиг. 1. A) Локализация PS811 (SEQ ID NO: 1673), PS814 (SEQ ID NO: 1675), PS815 (SEQ ID NO: 1677) и PS816 (SEQ ID NO: 1679) в участке последовательности, который является высоко сходным (63%) в экзоне 10 и экзоне 18. B) Характеристики и эффективность AON. Указан процент обратной комплементарности (обр. компл.) каждого AON в отношении либо экзона 10, либо экзона 18. C) Анализ способом ОТ-ПЦР. В мышечных клетках здорового человека (т.е. дифференцированных мышечных трубочках) все четыре AON были эффективными в отношении индукции пропускания мультиэкзонного участка с 10 экзона по 18 экзон. PS811 и PS816 были наиболее эффективными. В рамках слева на фигуре представлено содержание амплифицированных способом ПЦР продуктов транскриптов. (M: маркер размера ДНК; NT: не обработанные клетки).
Фиг. 2. A) Анализ ОТ-ПЦР. В мышечных клетках здорового человека (т.е. дифференцированных мышечных трубочках) исследовали AON с различной длиной, однако с одинаковой центральной последовательностью-мишенью в участке последовательности, который является высоко сходным (63%) в экзоне 10 и экзоне 18. PS816 (SEQ ID NO: 1679), 25-мер, и PS1059 (SEQ ID NO: 1684), 21-мер, были наиболее эффективными (пропускание экзонов 10 и 18 составляет 68% и 79%, соответственно). 15-мер PS1050 (SEQ ID NO: 1682) был менее эффективным. В рамках слева на фигуре представлено содержание амплифицированных способом ПЦР продуктов-транскриптов. (M: маркер размера ДНК; NT: необработанные клетки). B) Характеристики и эффективность AON. Указан процент обратной комплементарности (обр. компл.) каждого AON в отношении либо экзона 10, либо экзона 18.
Фигура 3. A) Анализ ОТ-ПЦР. В мышечных клетках здорового человека (т.е. дифференцированные мышечные трубочки) исследовали AON с различной химической структурой оснований: PS816 (AON с регулярной 2'-O-метилфосфоротиоатной РНК; SEQ ID NO: 1679), PS1168 (PS816, но в котором все As заменены на 2,6-диаминопурин; SEQ ID NO: 1681), PS1059 (AON с регулярной 2'-O-метилфосфоротиоатной РНК; SEQ ID NO: 1684), PS1138 (PS1059, но в котором все Cs заменены на 5-метилцитозины; SEQ ID NO: 1685) или PS1170 (PS1059, но в котором все As заменены на 2,6-диаминопурины; SEQ ID NO: 1686). Пропускание экзонов с 10 по 18 наблюдали для всех исследованных AON. PS816 был наиболее эффективным (88%). В этих конкретных последовательностях модификации оснований не повышали далее биоактивность. В рамках слева на фигуре указаны амплифицированные способом ПЦР продукты-транскрипты. (M: маркер размера ДНК; NT: не обработанные клетки). B) Характеристики и эффективность AON.
Фиг. 4. A) Высоко сходные участки последовательности в экзонах 10, 18, 30 и 47 в качестве множественных мишеней для PS816. SEQ ID NO относятся к полноразмерному выравниванию экзонов EMBOSS (как в таблице 2). Серым цветом выделена часть последовательности, которая не была включена в выравнивание EMBOSS, но которая является соседней с частью с высокой обратной комплементарностью с PS816. B) Обзор выравнивания экзонов и процентов обратной комплементарности (обр. компл.) с PS816. C) Анализ ОТ-ПЦР. С помощью PS816 может быть индуцировано пропускание мультиэкзонного участка, обозначаемое в настоящем описание как пропускание экзонов с 10 по 18, экзонов с 10 по 30 и экзонов с 10 по 47. Полученные транскрипты находятся в рамке считывания. Они не были выявлены в необработанных клетках (NT). В рамках слева и справа на фигуре представлено содержание амплифицированных способом ПЦР продуктов транскриптов. (M: маркер размера ДНК).
Фиг. 5. A) Локализация PS1185 (SEQ ID NO: 1706), PS1186 (SEQ ID NO: 1707) и PS1188 (SEQ ID NO: 1713) в участке последовательности, который является высоко сходным (80%) в экзоне 45 и экзоне 55. В таблице обобщенно представлены характеристики AON. Указан процент обратной комплементарности (обр. компл.) каждого AON в отношении либо экзона 45, либо экзона 55. B) Анализ ОТ-ПЦР. В мышечных клетках здорового человека (т.е. дифференцированные мышечные трубочки) все три AON были эффективными в отношении индукции пропускания мультиэкзонного участка с 45 по 55 экзоны. В рамках слева на фигуре представлено содержание амплифицированных способом ПЦР продуктов транскриптов. (M: маркер размера ДНК; NT: необработанные клетки).
Список ссылок
Aartsma-Rus A, Bremmer-Bout M, Janson AA, den Dunnen JT, van Ommen GJ, van Deutekom JC. Targeted exon skipping as a potential gene correction therapy for Duchenne muscular dystrophy. See 1 article found using an alternative search: Neuromuscul Disord. 2002; 12:S71-7.
Aartsma-Rus A, Janson AA, Kaman WE, Bremmer-Bout M, den Dunnen JT, Baas F, van Ommen GJ, van Deutekom JC. Therapeutic antisense-induced exon skipping in cultured muscle cells from six different DMD patients. Hum Mol Genet. 2003; 12(8):907-14.
Aartsma-Rus A, Janson AA, Kaman WE, Bremmer-Bout M, van Ommen GJ, den Dunnen JT, van Deutekom JC. Antisense-induced multiexon skipping for Duchenne muscular dystrophy makes more sense. Am. J. Hum. Genet. 2004; Jan 74(1):83-92.
Alloza I et al., Genes Immun 2012; 13(3):253-257.
Abeliovich A et al., J Neurochem 2006; 99:1062-1072.
van Ommen GJ, van Deutekom J, Aartsma-Rus A. The therapeutic potential of antisense-mediated exon skipping. Curr Opin Mol Ther. 2008; 10(2):140-9.
Andrade MA, Perez-Iratxeta C, Ponting CP. Protein repeats: structures, functions, and evolution. J Struct Biol. 2001; 134(2-3):117-31.
Arai K, Uchiyama N, Wada T. Synthesis and properties of novel 2'-O-alkoxymethyl-modified nucleic acids. Bioorg Med Chem Lett. 2011; 21(21):6285-7.
Béroud C, Tuffery-Giraud S, Matsuo M, Hamroun D, Humbertclaude V, Monnier N, Moizard MP, Voelckel MA, Calemard LM, Boisseau P, Blayau M, Philippe C, Cossée M, Pagès M, Rivier F, Danos O, Garcia L, Claustres M. Multiexon skipping leading to an artificial DMD protein lacking amino acids from exons 45 through 55 could rescue up to 63% of patients with Duchenne muscular dystrophy. Hum Mutat. 2007; Feb;28(2):196-202.
Bomont P et al., Nat Genet 2000; 26(3):370-374.
Cartegni L, Chew SL, Krainer AR. Listening to silence and understanding nonsense: exonic mutations that affect splicing. Nat Rev Genet 2002; 3(4):285-98.
Cartegni L, Wang J, Zhu Z, Zhang MQ, Krainer AR. ESEfinder: A web resource to identify exonic splicing enhancers. Nucleic Acids Res 2003; 31(13):3568-71.
Chabriat H et al., Lancet Neurol 2009; 8(7):643-653.
Cheng AJ and Van Dyke MW. Oligodeoxyribonucleotide length and sequence effects on intramolecular and intermolecular G-quartet formation. Gene. 1997; 197(l-2):253-60.
Cirak S, Arechavala-Gomeza V, Guglieri M, Feng L, Torelli S, Anthony K, Abbs S, Garralda ME, Bourke J, Wells DJ, Dickson G, Wood MJ, Wilton SD, Straub V, Kole R, Shrewsbury SB, Sewry C, Morgan JE, Bushby K, Muntoni F. Exon skipping and dystrophin restoration in patients with Duchenne muscular dystrophy after systemic phosphorodiamidate morpholino oligomer treatment: an open-label, phase 2, dose-escalation study. Lancet. 2011; 378(9791):595-605.
Cirak S et al., Brain 2010; 133(Pt 7):2123-2135.
Diebold SS, Massacrier C, Akira S, Paturel C, Morel Y, Reis e Sousa C. Nucleic acid agonists for Toll-like receptor 7 are defined by the presence of uridine ribonucleotides. Eur J Immunol; 2006; 36(12):3256-67.
Dhanoa BS et al., Hum Genomics 2013; 7(1):13.
Dorn A, Kippenberger S. Clinical application of CpG-, non-CpG-, and antisense oligodeoxynucleotides as immunomodulators. Curr Opin Mol Ther. 2008; 10(1):10-20.
Dubosq-Bidot L et al., Eur Heart J 2009; 30(17):2128-2136.
Ehmsen J. et al., J. Cell Sci. 2002, 115 (Pt14):2801-2803.
Fiorillo C et al., Neurogenetics 2012; 13(3):195-203.
Friedman JS et al., Am J Hum genet 2009; 84(6):792-800.
Goemans NM, Tulinius M, van den Akker JT, Burm BE, Ekhart PF, Heuvelmans N, Holling T, Janson AA, Platenburg GJ, Sipkens JA, Sitsen JM, Aartsma-Rus A, van Ommen GJ, Buyse G, Darin N, Verschuuren JJ, Campion GV, de Kimpe SJ, van Deutekom JC. Systemic administration of PRO051 in Duchenne's muscular dystrophy. N Engl J Med. 2011; 364(16):1513-22.
Goyenvalle A, Wright J, Babbs A, Wilkins V, Garcia L, Davies KE. Engineering Multiple U7snRNA Constructs to Induce Single and Multiexon-skipping for Duchenne Muscular Dystrophy. Mol Ther. 2012 Jun; 20(6):1212-21.
Heemskerk H, de Winter C, van Kuik P, Heuvelmans N, Sabatelli P, Rimessi P, Braghetta P, van Ommen GJ, de Kimpe S, Ferlini A, Aartsma-Rus A, van Deutekom JC. Preclinical PK and PD studies on 2'-O-methyl-phosphorothioate RNA antisense oligonucleotides in the mdx mouse model. Mol Ther. 2010; 18(6):1210-7.
Heneka MT et al., Nature2013; 493(7434):674-678.
Hodgetts S, Radley H, Davies M, Grounds MD. Reduced necrosis of dystrophic muscle by depletion of host neutrophils, or blocking TNFalpha function with Etanercept in mdx mice. Neuromuscul Disord. 2006; 16(9-10):591-602.
Hovnanian A, Cell Tissue Res 2013; 351(2):289-300.
Huang JY et al., Hum Reprod 2013; 28(4):1127-1134.
Knauf F et al., Kidney Int 2013; doi:10.1038/ki.2013.207.
Krieg AM, Yi AK, Matson S, Waldschmidt TJ, Bishop GA, Teasdale R, Koretzky GA, Klinman DM. CpG motifs in bacterial DNA trigger direct B-cell activation. Nature. 1995; 374(6522):546-9.
Krieg, AM. The role of CpG motifs in innate immunity. Curr. Opin. Immunol. 2000; 12:35-43.
Kumar L, Pharm. Technol. 2008, 3, 128.
Louis-Dit-Picard H et al., Nat Genet 2012; 44(4):456-460.
Macaya RF, Waldron JA, Beutel BA, Gao H, Joesten ME, Yang M, Patel R, Bertelsen AH, Cook AF. Structural and functional characterization of potent antithrombotic oligonucleotides possessing both quadruplex and duplex motifs. Biochemistry. 1995; 34(13):4478-92.
Manzur AY, Kuntzer T, Pike M, Swan A. Glucocorticoid corticosteroids for Duchenne muscular dystrophy. Cochrane Database Syst Rev. 2008; (1):CD003725.
Monaco A.P., et al., Genomics 1988; 2:90-95.
Peacock H, Fucini RV, Jayalath P, Ibarra-Soza JM, Haringsma HJ, Flanagan WM, Willingham A, Beal PA. Nucleobase and ribose modifications control immunostimulation by a microRNA-122-mimetic RNA. J. Am. Chem. Soc. 2011, 133(24):9200-3.
Mollie A et al., Hum Genet 2011; 129(6):641-654.
Moller RS et al., Epilepsia 2013; 54(2):256-264.
Noris P et al., Blood 2011; 117(24):6673-6680.
Popovic PJ, DeMarco R, Lotze MT, Winikoff SE, Bartlett DL, Krieg AM, Guo ZS, Brown CK, Tracey KJ, Zeh HJ 3rd. High mobility group B1 protein suppresses the human plasmacytoid dendritic cell response to TLR9 agonists. J of Immunol 2006; 177:8701-8707.
Puente XS et al., Nature 2011; 475(7354):101-105.
Raciti GA et al., Diabetologia 2011; 54(11):2911-2922.
Rantamaki T et al., Am J Hum Genet 1999; 64(4):993-1001.
Remington: The Science and Practice of Pharmacy, 20th Edition. Baltimore, MD: Lippincott Williams & Wilkins, 2000.
Sirmaci A et al., Am J Hum Genet 2011; 89(2):289-294.
Stuart HM et al., Am J Hum Genet 2013; 92(2):259-264.
Suzuki K, Doi T, Imanishi T, Kodama T, Tanaka T. Oligonucleotide aggregates bind to the macrophage scavenger receptor. Eur J Biochem. 1999; 260(3):855-60.
Uchida K et al., Immunology 2005; 116(1):53-63.
van Deutekom JC, Janson AA, Ginjaar IB, Frankhuizen WS, Aartsma-Rus A, Bremmer-Bout M, den Dunnen JT, Koop K, van der Kooi AJ, Goemans NM, de Kimpe SJ, Ekhart PF, Venneker EH, Platenburg GJ, Verschuuren JJ, van Ommen GJ. Local dystrophin restoration with antisense oligonucleotide PRO051. N Engl J Med. 2007; 357(26):2677-86.
van Vliet L, de Winter CL, van Deutekom JC, van Ommen GJ, Aartsma-Rus A. Assessment of the feasibility of exon 45-55 multiexon skipping for Duchenne muscular dystrophy. BMC Med Genet. 2008, Dec 1; 9:105.
Wagner, H., Bacterial CpG DNA activates immune cells to signal infectious danger Adv. Immunol. 1999; 73: 329-368.
Yokota T, Duddy W, Partridge T. Optimizing exon skipping therapies for DMD. Acta Myol. 2007; 26(3):179-84.
Yokota T, Hoffman E, Takeda S. Antisense oligo-mediated multiple exon skipping in a dog model of duchenne muscular dystrophy. Methods Mol Biol. 2011; 709:299-312.
Zuker M. Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Res. 2003; 31(13), 3406-3415.
| название | год | авторы | номер документа |
|---|---|---|---|
| ОЛИГОНУКЛЕОТИД ДЛЯ ЛЕЧЕНИЯ ПАЦИЕНТОВ С МЫШЕЧНОЙ ДИСТРОФИЕЙ | 2013 |
|
RU2789279C2 |
| Система направленного изменения сплайсинга в гене MARK2 | 2023 |
|
RU2810907C1 |
| АНТИСМЫСЛОВЫЕ НУКЛЕИНОВЫЕ КИСЛОТЫ | 2011 |
|
RU2567664C2 |
| МОДУЛЯТОРЫ И МОДУЛЯЦИЯ РНК РЕЦЕПТОРА КОНЕЧНЫХ ПРОДУКТОВ ГЛУБОКОГО ГЛИКИРОВАНИЯ | 2020 |
|
RU2820247C2 |
| АНТИСМЫСЛОВЫЕ НУКЛЕИНОВЫЕ КИСЛОТЫ | 2012 |
|
RU2619184C2 |
| АНТИСМЫСЛОВЫЕ НУКЛЕИНОВЫЕ КИСЛОТЫ | 2018 |
|
RU2681470C1 |
| АНТИСМЫСЛОВЫЕ НУКЛЕИНОВЫЕ КИСЛОТЫ | 2012 |
|
RU2651468C1 |
| АНТИСМЫСЛОВЫЕ НУКЛЕИНОВЫЕ КИСЛОТЫ | 2015 |
|
RU2702424C2 |
| АНТИСМЫСЛОВЫЕ НУКЛЕИНОВЫЕ КИСЛОТЫ | 2015 |
|
RU2730681C2 |
| НОВЫЙ СПОСОБ ЛЕЧЕНИЯ ПИГМЕНТНОГО РЕТИНИТА | 2020 |
|
RU2817702C2 |




































































































































































































































































































































































































































































































































































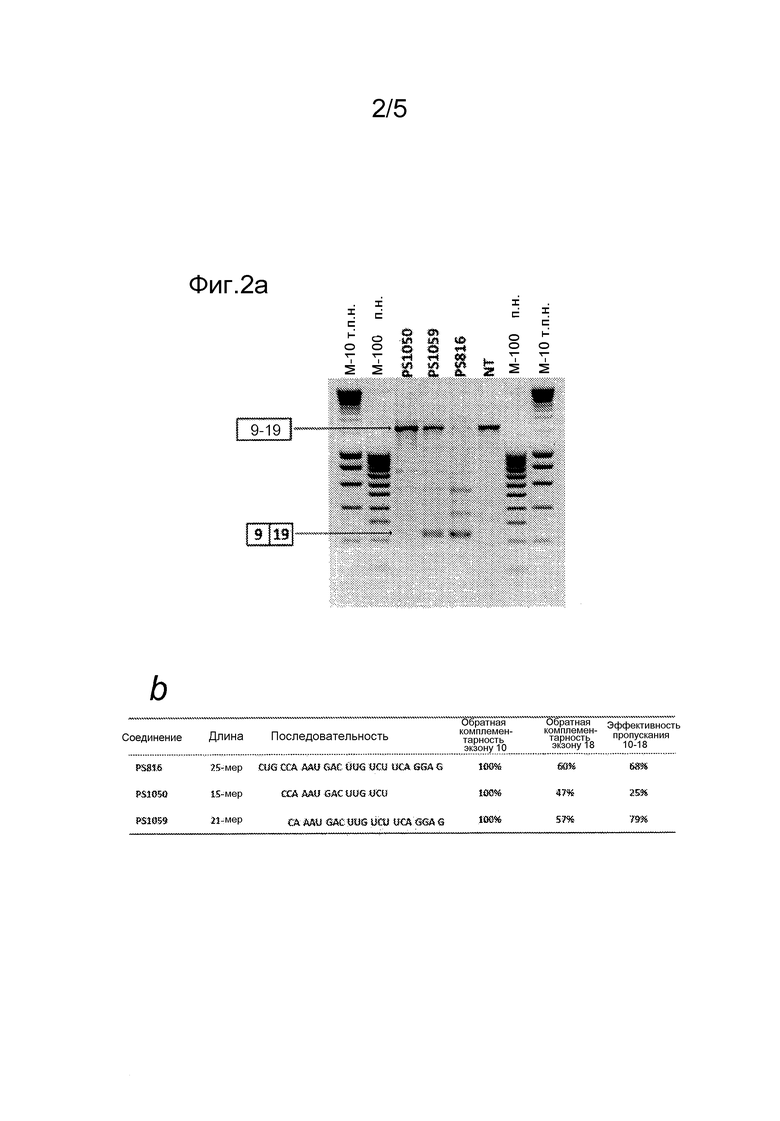



Данное изобретение относится к биотехнологии. Предложен олигонуклеотид для обеспечения пропуска двух или более экзонов пре-мРНК дистрофина. Также рассмотрена композиция для обеспечения пропуска двух или более экзонов пре-мРНК дистрофина, способ и применение олигонуклеотида и композиции для предупреждения, замедления, смягчения и/или лечения мышечной дистрофии Дюшенна (DMD) и мышечной дистрофии Беккера (BMD). Настоящее изобретение может найти дальнейшее применение в терапии различных заболеваний, вызванных мутациями в гене дистрофина. 4 н. и 1 з.п. ф-лы, 5 ил., 6 табл., 5 пр.
1. Олигонуклеотид для обеспечения пропуска двух или более экзонов пре-мРНК дистрофина, содержащий последовательность оснований или ее часть, как определено в SEQ ID NO: 1679, и имеющий длину 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов, где указанная часть соответствует последовательности оснований, меньшей на 1, 2, 3, 4 или 5 нуклеотидов, чем определено в указанной последовательности оснований.
2. Композиция для обеспечения пропуска двух или более экзонов пре-мРНК дистрофина, содержащая олигонуклеотид по п.1 и необязательно дополнительно содержащая фармацевтически приемлемый носитель, разбавитель, эксципиент, соль, адъювант и/или растворитель, где концентрация олигонуклеотидов в композиции находится в пределах от 0,02 до 400 нМ.
3. Композиция по п.2, где указанная композиция содержит два или более идентичных олигонуклеотидов, как определено в п.1, связанных противоионом, предпочтительно кальцием.
4. Способ предупреждения, замедления, смягчения и/или лечения мышечной дистрофии Дюшенна (DMD) или мышечной дистрофии Беккера (BMD) у индивидуума, причем указанный способ включает введение указанному индивидууму олигонуклеотида по п.1 или композиции по п.2 или 3.
5. Применение олигонуклеотида по п.1 или композиции по п.2 или 3 для предупреждения, замедления, смягчения и/или лечения BMD или DMD.
| Способ приготовления мыла | 1923 |
|
SU2004A1 |
| Способ приготовления лака | 1924 |
|
SU2011A1 |
| Пломбировальные щипцы | 1923 |
|
SU2006A1 |
| Способ восстановления хромовой кислоты, в частности для получения хромовых квасцов | 1921 |
|
SU7A1 |
| Приспособление для суммирования отрезков прямых линий | 1923 |
|
SU2010A1 |
| AARTSMA-RUS A | |||
| et al | |||
| "Antisense-induced multiexon skipping for Duchenne muscular dystrophy makes more sense." The American Journal of Human Genetics, 2004; 74(1): 83-92. | |||
Авторы
Даты
2018-12-11—Публикация
2013-07-03—Подача