Данная заявка испрашивает приоритет на основании заявки на патент США с серийным номером 61/777238, поданной 12 марта 2013 года, которая включена в настоящий документ посредством ссылки во всей своей полноте.
ОБЛАСТЬ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к области молекулярной биологии. Более конкретно, настоящее изобретение относится к способам идентификации и применения вариантных сайтов распознавания для редкощепящих сконструированных средств для индукции двунитевого разрыва.
ПРЕДПОСЫЛКИ ИЗОБРЕТЕНИЯ
Технология рекомбинантных ДНК сделала возможным встраивание чужеродных последовательностей ДНК в геном организма, таким образом, изменяя фенотип организма. Наиболее широко используемые способы трансформации растений представляют собой инфицирование с помощью Agrobacterium и бомбардировку частицами в ходе биолистической трансформации, при которой трансгены интегрируются в геном растения случайным образом и с непрогнозируемым числом копий. Таким образом, предпринимают попытки контроля интеграции трансгенов в растения.
Были разработаны способы введения или модификации последовательности ДНК в геноме ряда организмов, и они могут включать методики сайт-специфической интеграции, которые основываются на гомологичной рекомбинации (патент США № 7102055, выданный 05 сентября 2006 года) или дизайнерских эндонуклеазах, таких как мегануклеазы, нуклеазы "цинковые пальцы" или TALEN (публикация патента США 2009-0133152 A1, опубликованного 21 мая 2009 года).
Хотя эти системы обеспечили полезные методики для целенаправленной вставки последовательностей, представляющих интерес, остается потребность в идентификации большего количества сайтов распознавания для редкощепящих средств для индукции двунитевого разрыва и в идентификации сайтов распознавания с повышенной активностью в отношении редкощепящих средств для индукции двунитевого разрыва.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Предусматриваются композиции и способы, в которых используются вариантные сайты распознавания для редкощепящего сконструированного средства для индукции двунитевого разрыва, способного к введению редкого двунитевого разрыва в предполагаемом сайте распознавания.
Предусматриваются способы идентификации вариантного сайта распознавания для редкощепящего сконструированного средства для индукции двунитевого разрыва, способного к введению редкого двунитевого разрыва в предполагаемом сайте распознавания. Один способ включает a) приведение в контакт геномной ДНК с редкощепящим сконструированным средством для индукции двунитевого разрыва, способным к введению двунитевого разрыва в указанную геномную ДНК, где двунитевой разрыв приводит в результате к образованию нуклеотидного липкого конца, b) лигирование первого адаптера с указанным нуклеотидным липким концом, c) разрезание лигированной ДНК, полученной на стадии (b), и лигирование по меньшей мере одного второго адаптера с разрезанным нуклеотидным концом для обеспечения амплификации и секвенирования фрагментов геномной ДНК, окружающих двунитевой разрыв, d) выравнивание нуклеотидных последовательностей фрагментов ДНК, полученных в (c), с эталонной последовательностью геномной ДНК; и e) идентификацию вариантного сайта распознавания, содержащего изменение по меньшей мере по одному нуклеотидному основанию по сравнению с предполагаемым сайтом распознавания указанного сконструированного средства для индукции двунитевого разрыва. Другой способ включает способ идентификации вариантного сайта распознавания для редкощепящего сконструированного средства для индукции двунитевого разрыва, способного к введению редких двунитевых разрывов в предполагаемый сайт распознавания, причем указанный способ включает: a) приведение в контакт геномной ДНК с редкощепящим сконструированным средством для индукции двунитевого разрыва, способным к введению двунитевого разрыва в указанную геномную ДНК, где двунитевой разрыв приводит в результате к образованию тупого конца; b) создание нуклеотидного липкого конца из тупого конца из (a); c) лигирование первого адаптера с нуклеотидным липким концом из (b); d) разрезание лигированной ДНК, полученной на стадии (c) и лигирование по меньшей мере одного второго адаптера с разрезанным нуклеотидным концом для обеспечения амплификации и секвенирования фрагментов геномной ДНК, окружающих двунитевой разрыв; e) выравнивание нуклеотидных последовательностей фрагментов ДНК, полученных в (d), с эталонной геномной последовательностью ДНК и f) идентификацию вариантного сайта распознавания, содержащего изменение по меньшей мере по одному нуклеотидному основанию по сравнению с предполагаемым сайтом распознавания указанного сконструированного средства для индукции двунитевого разрыва.
Редкощепящее сконструированное средство для индукции двунитевого разрыва можно выбрать из группы, состоящей из мегануклеазы, нуклеазы "цинковые пальцы", TAL-эффекторной нуклеазы, транспозазы, эндонуклеазы Cas и сайт-специфической рекомбиназы. Нуклеотидный липкий конец может представлять собой 3'- или 5'-нуклеотидный липкий конец.
Кроме того предусматриваются способы идентификации вариантного сайта распознавания с улучшенной активностью расщепления для редкощепящего сконструированного средства для индукции двунитевого разрыва, способного к введению двунитевого разрыва в предполагаемый сайт распознавания. О повышенной активности редкощепящего сконструированного средства для индукции двунитевого разрыва свидетельствуют a) более высокий процент (%) расщепления вариантного сайта распознавания по сравнению с процентом (%) расщепления предполагаемого сайта распознавания, где сайты распознавания расположена на геномной ДНК; b) более высокий процент (%) расщепления вариантного сайта распознавания по сравнению с процентом (%) расщепления предполагаемого сайта распознавания, где сайты распознавания расположены на плазмидной ДНК; c) более высокая оценка в анализе на дрожжах для вариантного сайта распознавания по сравнению с предполагаемым сайтом распознавания; или d) любая комбинация из (a), (b) и (c).
Также предусматриваются способы нацеливания вставки полинуклеотида, представляющего интерес, на конкретный хромосомный сайт в геноме растения, причем указанный способ включает: a) трансформацию растительной клетки или растения фрагментом ДНК, содержащим полинуклеотид, представляющий интерес, где указанный геном указанных растительной клетки или растения содержит по меньшей мере один вариантный сайт распознавания, выбранный из группы, состоящей из SEQ ID NO: 15, 16, 17, 18, 19, 20 и 21 или SEQ ID NO: 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34 и 35; и b) обеспечение наличия мегануклеазы, способной к обеспечению двунитевого разрыва в вариабельном сайте распознавания из (a); и c) отбор указанных растительной клетки или растения, содержащих указанный полинуклеотид, представляющий интерес, интегрированный в указанный вариантный сайт распознавания.
Различные композиции включают растение, семя или растительную клетку, содержащую в своем геноме вариантный сайт распознавания для редкощепящего сконструированного средства для индукции двунитевого разрыва, способного к введению редкого двунитевого разрыва в предполагаемый сайт распознавания.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ И ПЕРЕЧНЯ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
Настоящее изобретение может быть понятным в более полном объеме из следующего подробного описания и прилагаемых графических материалов и перечня последовательностей, которые образуют часть настоящей заявки. Описания последовательностей и перечень последовательностей, включенные в качестве приложения в данный документ, соответствуют правилам, регулирующим раскрытия нуклеотидных и аминокислотных последовательностей в патентных заявках, как изложено в §1.821-1.825 раздела 37 C.F.R. Описания последовательностей содержат трехбуквенные коды аминокислот, как определено в §§1.821-1.825 раздела 37 C.F.R., которые включены в данный документ посредством ссылки.
Фигура 1. (A) Сигнатура пика для геномного сайта распознавания из образца, обработанного мегануклеазой. Нанесенные на карту данные последовательности начинаются с сайта расщепления и возвращаются к нему. Направление нанесенных на карту ридов указывается в виде с наложением и показано стрелками. (B) Пустой контроль не содержит сигнатуры, соответствующей обогащению или пику, как наблюдалось для обработанного образца.
Фигура 2. Перекрытие 4 оснований в сигнатуре пика сайта распознавания соответствует липкому концу, созданному мегануклеазой, и определяет последовательность геномного вариантного сайта распознавания. Пунктирная линия определяет липкие концы, полученные при расщеплении сайта распознавания.
Фигура 3. Процентный состав оснований в ДНК ориентированных геномных вариантных сайтов распознавания. Предпочтительные посторонние нуклеотиды обведены, тогда как основания предполагаемого сайта распознавания заштрихованы. (A) Состав оснований ДНК 30 геномных вариантных сайтов распознавания мегануклеазой Lig3-4. Предполагаемый сайт распознавания LIG3-4 (SEQ ID NO:13) показан снизу фигуры 3A. (B) Состав оснований ДНК 254 геномных вариантных сайтов распознавания мегануклеазой MHP14+. Предполагаемый сайт распознавания MHP14+ (SEQ ID NO:14) показан снизу фигуры 3B.
Фигура 4. A) Выравнивание предполагаемого сайта распознавания для мегануклеазы LIG3-4 и вариантных сайтов распознавания LIG3-4. Предпочтительные посторонние нуклеотиды (подчеркнутые) вводили в предполагаемый сайт распознавания LIG3-4 по отдельности (что приводило к созданию вариантных сайтов распознавания -11C, -7C, -2G, -1T, +8T, соответствующих SEQ ID NO: 15-19) и в комбинации (что приводило к созданию вариантных сайтов распознавания, соответствующих SEQ ID NO:20-22). B) Выравнивание предполагаемого сайта распознавания для мегануклеазы MHP14+ и вариантных сайтов распознавания MHP14+. Предпочтительные посторонние нуклеотиды (подчеркнутые) вводили в предполагаемый сайт распознавания MHP14+ по отдельности (что приводило к созданию вариантных сайтов распознавания -3A, -2G, -1T, +2A, +7T, +8G, +11G, +11A, соответствующих SEQ ID NO: 23-30) и в комбинации (что приводило к созданию вариантных сайтов распознавания, соответствующих SEQ ID NO: 31-35).
Фигура 5. Сравнение активности расщепления плазмидной ДНК между предполагаемым сайтом распознавания и предполагаемыми посторонними нуклеотидами, помещенными по отдельности в предполагаемый сайт распознавания для (A) мегануклеазы Lig3-4 и (B) мегануклеазы MHP14+. Процент активности расщепления предполагаемого сайта распознавания помечен пунктирной линией. В качестве контроля также анализировали по меньшей мере два основания, не предпочтительных по процентному составу оснований ДНК в геномных вариантных сайтах распознавания.
Фигура 6. Сравнение активности расщепления плазмидной ДНК между предполагаемым сайтом распознавания и предпочтительными посторонними нуклеотидами, помещенными в комбинации в предполагаемый сайт распознавания для (A) мегануклеазы Lig3-4 и (B) мегануклеазы MHP14+.
Фигура 7. Сравнение графиков амплификации ПЦР в реальном времени из библиотек геномных вариантных сайтов распознавания для мегануклеазы, созданных либо с фосфорилированными, либо с нефосфорилированными биотинилированными адаптерами, содержащими полностью вырожденный 4-нуклеотидный 3'-липкий конец.
На фигуре 8 показана диаграмма, представляющая систему скрининга дрожжей, применяемую для определения мегануклеазной активности у дрожжей. Фрагменты гена, соответствующие первым 1000 нуклеотидам кодирующей последовательности Ade2 дрожжей (5’-фрагмент Ade2) и последним 1011 нуклеотидам кодирующей последовательности Ade2 дрожжей (3’-фрагмент Ade2), разъединяли с помощью фрагмента, содержащего ген дрожжей ura3 (Ura3) и сайты распознавания мегануклеазой для I-SceI.
На фигуре 9 показана числовая шкала и соответствующее образование белых секторов в колониях дрожжей, применяемых для количественного определения мегануклеазной активности. Поскольку фенотип образования секторов является качественным показателем мегануклеазной активности, задействовали числовую систему количественных показателей 0-4. Количественный показатель 0 означает, что белые сектора не наблюдались (отсутствовало разрезание мегануклеазами); количественный показатель 4 означает полностью белые колонии (полное разрезание сайта распознавания); количественные показатели 1-3 означают промежуточные фенотипы образования белых секторов (и промежуточные степени разрезания сайта распознавания).
Фигура 10. (A) Сравнение активности расщепления плазмидной ДНК между предполагаемым сайтом распознавания MHP14+ (SEQ ID NO: 14), вариантным сайтом распознавания MHP14+ (SEQ ID NO: 11), который встречается в естественных условиях в геноме маиса (меченный вариантный сайт распознавания маиса), и вариантными сайтами распознавания MHP14+ (SEQ ID NO: 31-35), которые не являются эндогенными для генома маиса. (B) Сравнение относительного числа копий вариантного сайта распознавания MHP14+ с SEQ ID NO:11 и предполагаемого сайта распознавания MHP14+ (SEQ ID NO: 14) в зрелых зародышах маиса.
Фигура 11. (A) представляет собой карту плазмиды PHP57712, (B) представляет собой карту плазмиды PHP62552.
ПОСЛЕДОВАТЕЛЬНОСТИ
SEQ ID NO: 1 представляет собой нуклеотидную последовательность, кодирующую одноцепочечный полипептид слияния на основе мегануклеазы LIG3-4.
SEQ ID NO: 2 представляет собой аминокислотную последовательность полипептида слияния на основе мегануклеазы LIG3-4.
SEQ ID NO: 3 представляет собой нуклеотидную последовательность, кодирующую одноцепочечную мегануклеазу MHP14+.
SEQ ID NO: 4 представляет собой аминокислотную последовательность мегануклеазы MHP14+.
SEQ ID NO: 5 представляет собой нуклеотидную последовательность биотинилированного дефосфорилированного адаптера, сконструированного с полностью вырожденным 4-п.о. 3'-липким концом.
SEQ ID NO: 6 представляет собой нуклеотидную последовательность восстанавливающего праймера A.
SEQ ID NO: 7 представляет собой нуклеотидную последовательность восстанавливающего праймера B.
SEQ ID NO: 8 представляет собой нуклеотидную последовательность Illumina-совместимого адаптера.
SEQ ID NO: 9 представляет собой нуклеотидную последовательность маркерной последовательности.
SEQ ID NO: 10 представляет собой комплементарную нуклеотидную последовательность маркерной последовательности с SEQ ID NO: 9.
SEQ ID NO: 11 представляет собой нуклеотидную последовательность 5'-3' последовательности, показанной на фигуре 2.
SEQ ID NO: 12 представляет собой нуклеотидную последовательность 3'-5' последовательности, показанной на фигуре 2.
SEQ ID NO: 13 представляет собой нуклеотидную последовательность предполагаемого сайта распознавания для мегануклеазы LIG3-4 (также показана на фигуре 3A и фигуре 4).
SEQ ID NO: 14 представляет собой нуклеотидную последовательность предполагаемого сайта распознавания для мегануклеазы MHP14+ (также показана на фигуре 3B и фигуре 4).
SEQ ID NO:15-22 представляют собой нуклеотидные последовательности вариантных сайтов распознавания для мегануклеазы LIG3-4.
SEQ ID NO:23-36 представляют собой нуклеотидные последовательности вариантных сайтов распознавания для мегануклеазы MHP14+.
SEQ ID NO: 36 представляет собой нуклеотидную последовательность гена Ade2 дрожжей.
SEQ ID NO: 37 представляет собой нуклеотидную последовательность предполагаемого сайта распознавания для мегануклеазы MS26.
SEQ ID NO: 38 представляет собой нуклеотидную последовательность плазмиды PHP57712.
SEQ ID NO: 39 представляет собой нуклеотидную последовательность плазмиды PHP62552
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Настоящее изобретение теперь будет более подробно описано ниже в данном документе со ссылкой на прилагаемые графические материалы, на которых показаны некоторые, но не все варианты осуществления настоящего изобретения. В действительности, настоящее изобретение можно осуществлять во многих различных формах и его не следует рассматривать как ограниченное вариантами осуществления, изложенными в данном документе; скорее данные варианты осуществления представлены с тем, чтобы данное раскрытие соответствовало требованиям действующего законодательства. Одинаковые числа относятся к одинаковым элементам по всему документу.
Специалисту в данной области, к которой относится настоящее изобретение, придет на ум множество модификаций и других вариантов осуществления настоящего изобретения, изложенного в данном документе, при использовании идей, представленных в вышеприведенном описании и сопутствующих графических материалах. Таким образом, следует понимать, что настоящее изобретение не должно ограничиваться конкретными раскрытыми вариантами осуществления, и подразумевается, что в объем прилагаемой формулы изобретения включены модификации и другие варианты осуществления. Хотя в данном документе используются специальные выражения, они используются только в общем и описательном смысле, а не в целях ограничения.
Все публикации и заявки на патент, упомянутые в настоящем описании, ориентированы на уровень специалиста в данной области, к которой относится настоящее изобретение. Все публикации и заявки на патент включены в данный документ посредством ссылки в той же степени, как если бы каждая отдельная публикация или заявка на патент конкретно и отдельно была включена посредством ссылки.
Используемые в данном документе и в прилагаемой формуле изобретения формы единственного числа включают ссылки на множественное число, если из контекста явно не следует иное. Таким образом, например, ссылка на "растение" включает в себя множество таких растений; ссылка на "клетку" включает в себя одну или несколько клеток и их эквиваленты, известные специалистам в данной области техники, и так далее.
Если не указано иное, все технические и научные термины, применяемые в данном документе, имеют такое же значение, которое обычно понимается средним специалистом в области техники, к которой принадлежит настоящее изобретение. Хотя любые способы и материалы, подобные или эквивалентные описанным в данном документе, можно применять на практике для тестирования настоящего изобретения, в данном документе описаны конкретные примеры подходящих материалов и способов.
В контексте данного раскрытия используется ряд терминов и сокращений. Предлагаются следующие определения.
Как используется в данном документе, термины "целевой сайт", "целевая последовательность", "геномный целевой сайт" и "геномная целевая последовательность" используются в данном документе взаимозаменяемо и относятся к последовательности полинуклеотида в геноме растительной клетки или дрожжевой клетки, которая содержит сайт распознавания для средства для индукции двунитевого разрыва.
"Искусственный целевой сайт" представляет собой целевую последовательность, которая была введена в геном организма, такого как растение или дрожжи. Такая искусственная целевая последовательность может быть идентичной по последовательности по отношению к эндогенной или нативной целевой последовательности в геноме организма, но может располагаться в другом положении (т.е. неэндогенном или ненативном положении) в геноме организма.
Термины "эндогенная целевая последовательность" и "нативная целевая последовательность" используются взаимозаменяемо в данном документе для обозначения целевой последовательности, которая является эндогенной или нативной по отношению к геному хозяина (такого как растение или дрожжи) и и находится в эндогенном или нативном положении этой целевой последовательности в геноме хозяина (такого как растение или дрожжи).
Используемый в данном документе термин "средство для индукции двунитевого разрыва" относится к любой нуклеазе, которая производит двунитевой разрыв в целевой последовательности. Получение двунитевого разрыва в целевой последовательности или другой ДНК можно назвать в данном документе "разрезанием" или "расщеплением" целевой последовательности или другой ДНК.
Используемый в данном документе термин "редкощепящее средство для индукции двунитевого разрыва" относится к любой нуклеазе, которая производит двунитевой разрыв в целевой последовательности, но в редких случаях режет (в отличие от рестрикционных ферментов, например) в геноме организма. Редкощепящие средства для индукции двунитевого разрыва включают без ограничения эндонуклеазы, такие как мегануклеазы (заявка на патент США 2332 и BB1990), нуклеазы "цинковые пальцы" (Kim, Y. G., J. Cha, et al. (1996). "Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage), эндонуклеазы Cas (заявка WO2007/025097, опубликованная 1 марта 2007 года) и TALEN (Christian, M., T. Cermak, et al. 2010. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics 186(2): 757-61). Расщепление редкощепящими эндонуклеазами обычно приводит к образованию "липких" концов с 3'-липкими концами для мегануклеаз LAGLIDADG (Chevalier, B. S. and B. L. Stoddard. 2001. Homing endonucleases: structural and functional insight into the catalysts of intron/intein mobility. Nucleic Acids Res 29(18): 3757-74) и 5'-липкими концами для нуклеаз "цинковые пальцы" (Smith, J., M. Bibikova, et al. 2000, Requirements for double-strand cleavage by chimeric restriction enzymes with zinc finger DNA-recognition domains. Nucleic Acids Res 28(17): 3361-9). TALE-нуклеазы на основе Fokl (TALEN) характеризуются функциональной схемой, подобной нуклеазам "цинковые пальцы", причем ДНК-связывающий домен "цинковые пальцы" заменен на домен TALE (Li, T., S. Huang, et al. 2011. TAL nucleases (TALNs): hybrid proteins composed of TAL effectors and Fokl DNA-cleavage domain. Nucleic Acids Res 39(1): 359-72; Christian, M., T. Cermak, et al. 2010). Расщепление эндонуклеазами Cas, такими как эндонуклеазы Cas9, может приводить к тупым концам.
"Эндонуклеаза" относится к ферменту, который расщепляет фосфодиэфирную связь в цепи полинуклеотида.
Эндонуклеазы включают рестрикционные эндонуклеазы, расщепляющие ДНК в конкретных сайтах без повреждения оснований. Рестрикционные эндонуклеазы включают эндонуклеазы типа I, типа II, типа III и типа IV, которые дополнительно включают подтипы. В системах типа I и типа III ферментативные активности как метилазы, так и рестриктазы заключены в единый комплекс.
Рестрикционные эндонуклеазы типа I и типа III распознают специфические сайты распознавания, но, как правило, расщепляют в положении, отличном от сайта распознавания, которое может располагаться на расстоянии сотен пар оснований от сайта распознавания. В системах типа II рестрикционная активность не зависит от какой-либо метилазной активности, и расщепление обычно происходит в конкретных сайтах в пределах сайта распознавания или около него. Большинство ферментов типа II производят разрезание палиндромных последовательностей, однако ферменты типа IIa распознают непалиндромные сайты распознавания и производят расщепление за пределами сайта распознавания; ферменты типа IIb производят разрезание последовательностей дважды, причем оба сайта находятся за пределами сайта распознавания; и ферменты типа IIs распознают асимметричный сайт распознавания и производят расщепление с одной стороны и на определенном расстоянии от сайта распознавания, составляющем примерно 1-20 нуклеотидов. Ферменты рестрикции типа IV целенаправленно воздействуют на метилированную ДНК. Ферменты рестрикции дополнительно описаны и классифицированы, например, в базе данных REBASE (веб-страница на rebase.neb.com; Roberts et al., (2003) Nucleic Acids Res 31:418-20), Roberts et al., (2003) Nucleic Acids Res 31:1805-12; и Belfort et al., (2002) в Mobile DNA II, pp. 761-783, Eds. Craigie et al., (ASM Press, Washington, DC).
"Сконструированное редкощепящее средство для индукции двунитевого разрыва" относится к любому редкощепящему средству для индукции двунитевого разрыва, которое является сконструированным (модифицированным или происходящим) по сравнению с его нативной формой для специфического распознавания необходимого сайта распознавания и индукции двунитевого разрыва в нем. Таким образом, сконструированное редкощепящее средство для индукции двунитевого разрыва можно получить из нативной встречающейся в естественных условиях нуклеазы или его можно искусственно создать или синтезировать. Модификация нуклеазы может затрагивать всего лишь один нуклеотид. В некоторых вариантах осуществления сконструированное редкощепящее средство для индукции двунитевого разрыва индуцирует двунитевой разрыв в сайте распознавания, при этом сайт распознавания не являлся последовательностью, которая должна была быть распознанной нативным (несконструированным или немодифицированным) редкощепящим средством для индукции двунитевого разрыва. Получение двунитевого разрыва в сайте распознавания или другой ДНК можно назвать в данном документе "разрезанием" или "расщеплением" сайта распознавания или другой ДНК.
"Мегануклеаза" относится к хоминг-эндонуклеазе, которая, подобно рестрикционным эндонуклеазам, связывается с конкретным сайтом распознавания и разрезает его, однако сайты распознавания для мегануклеаз обычно являются большими, имея размер примерно 18 п.о. или более. В некоторых вариантах осуществления настоящего изобретения мегануклеаза была сконструирована (или модифицирована) для разрезания конкретной эндогенной последовательности распознавания, где эндогенная целевая последовательность перед разрезанием с помощью сконструированного средства, индуцирующего двунитевой разрыв, не была последовательностью, которая должна была быть распознанной нативной (несконструированной или немодифицированной) эндонуклеазой.
"Полипептид c мегануклеазной активностью" относится к полипептиду, характеризующемуся мегануклеазной активностью и, следовательно, способному производить двунитевой разрыв в распознаваемой последовательности.
Мегануклеазы были распределены в четыре семейства на основе консервативных мотивов последовательностей, при этом семейства представляют собой семейства LAGLIDADG, GIY-YIG, H-N-H и His-Cys-бокс. Эти мотивы участвуют в координации ионов металлов и гидролизе фосфодиэфирных связей. HEазы (HEases) примечательны своими длинными сайтами распознавания и тем, что допускают некоторые полиморфизмы последовательностей в их ДНК-субстратах. Правила номенклатуры мегануклеаз похожи на правила для других рестрикционных эндонуклеаз. Мегануклеазы также характеризуют по приставке F-, I- или PI- для ферментов, кодируемых автономными открытыми рамками считывания, интронами и интеинами, соответственно. Например, мегануклеазы от Saccharomyces cerevisiae, кодируемые интронами, интеинами и автономными генами, обозначаются I-SceI, PI-SceI и F-SceII, соответственно. Домены, структура и функции мегануклеаз известны; см., например, Guhan and Muniyappa (2003) Crit Rev Biochem Mol Biol 38:199-248; Lucas et al., (2001) Nucleic Acids Res 29:960-9; Jurica and Stoddard, (1999) Cell Mol Life Sci 55:1304-26; Stoddard, (2006) Q Rev Biophys 38:49-95; и Moure et al., (2002) Nat Struct Biol 9:764. В некоторых примерах применяют встречающийся в природе вариант и/или сконструированную мегануклеазу-производное. Известны способы модификации кинетических характеристик, взаимодействий с кофакторами, экспрессии, оптимальных условий и/или специфичности в отношении сайта распознавания и скрининга в отношении активности, см., например, Epinat et al., (2003) Nucleic Acids Res 31:2952-62; Chevalier et al., (2002) Mol Cell 10:895-905; Gimble et al., (2003) Mol Biol 334:993-1008; Seligman et al., (2002) Nucleic Acids Res 30:3870-9; Sussman et al., (2004) J Mol Biol 342:31-41; Rosen et al., (2006) Nucleic Acids Res 34:4791-800; Chames et al., (2005) Nucleic Acids Res 33:e178; Smith et al., (2006) Nucleic Acids Res 34:e149; Gruen et al., (2002) Nucleic Acids Res 30:e29; Chen and Zhao, (2005) Nucleic Acids Res 33:e154; WO2005105989; WO2003078619; WO2006097854; WO2006097853; WO2006097784 и WO2004031346.
В данном документе можно применять любую мегануклеазу, в том числе, без ограничений, I-SceI, I-SceII, I-SceIII, I-SceIV, I-SceV, I-SceVI, I-SceVII, I-CeuI, I-CeuAIIP, I-CreI, I-CrepsbIP, I-CrepsbIIP, I-CrepsbIIIP, I-CrepsbIVP, I-TliI, I-PpoI, PI-PspI, F-SceI, F-SceII, F-SuvI, F-TevI, F-TevII, I-AmaI, I-AniI, I-ChuI, I-CmoeI, I-CpaI, I-CpaII, I-CsmI, I-CvuI, I-CvuAIP, I-DdiI, I-DdiII, I-DirI, I-DmoI, I-HmuI, I-HmuII, I-HsNIP, I-LlaI, I-MsoI, I-NaaI, I-NanI, I-NcIIP, I-NgrIP, I-NitI, I-NjaI, I-Nsp236IP, I-PakI, I-PboIP, I-PcuIP, I-PcuAI, I-PcuVI, I-PgrIP, I-PobIP, I-PorI, I-PorIIP, I-PbpIP, I-SpBetaIP, I-ScaI, I-SexIP, I-SneIP, I-SpomI, I-SpomCP, I-SpomIP, I-SpomIIP, I-SquIP, I-Ssp6803I, I-SthPhiJP, I-SthPhiST3P, I-SthPhiSTe3bP, I-TdeIP, I-TevI, I-TevII, I-TevIII, I-UarAP, I-UarHGPAIP, I-UarHGPA13P, I-VinIP, I-ZbiIP, PI-MtuI, PI-MtuHIP, PI-MtuHIIP, PI-PfuI, PI-PfuII, PI-PkoI, PI-PkoII, PI-Rma43812IP, PI-SpBetaIP, PI-SceI, PI-TfuI, PI-TfuII, PI-ThyI, PI-TliI, PI-TliII, или любые их активные варианты или фрагменты. В конкретном варианте осуществления сконструированная эндонуклеаза происходит от I-Cre-I, имеющей последовательность, приведенную под SEQ ID NO: 15, 21 или 26, или ее активного варианта или фрагмента.
TAL-эффекторные нуклеазы являются новым классом нуклеаз, специфичных для последовательностей, которые можно применять для получения двунитевых разрывов в конкретных целевых последовательностях в геноме растения или другого организма. TAL-эффекторные нуклеазы создают путем слияния нативного или сконструированного эффектора, подобного активатору транскрипции (TAL), или функциональной части такового, с каталитическим доменом эндонуклеазы, такой как, например, FokI. Уникальный, модульный ДНК-связывающий домен TAL-эффектора создает возможность разработки белков с потенциальной специфичностью распознавания любой заданной ДНК. Таким образом, ДНК-связывающие домены TAL-эффекторных нуклеаз можно конструировать для распознавания конкретных целевых сайтов ДНК и, таким образом, применять для получения двунитевых разрывов в необходимых целевых последовательностях. См. WO 2010/079430; Morbitzer et al. (2010) PNAS 10.1073/pnas.1013133107; Scholze & Boch (2010) Virulence 1:428-432; Christian et al. Genetics (2010) 186:757-761; Li et al. (2010) Nuc. Acids Res. (2010) doi:10.1093/nar/gkq704; и Miller et al. (2011) Nature Biotechnology 29:143-148; все из которых включены в данный документ посредством ссылки.
Как используется в данном документе, выражение "ген Cas" относится к гену, который, как правило, соединен, связан или расположен вблизи или рядом с фланкирующими локусами CRISPR.
CRISPR-локусы (короткие палиндромные повторы, регулярно расположенные группами) (также известные как SPIDR - диспергированные повторы) составляют семейство недавно описанных локусов ДНК. CRISPR-локусы состоят из коротких и высококонсервативных повторов ДНК (обычно от 24 до 40 п.о., повторяющихся от 1 до 140 раз, также обозначаемых как CRISPR-повторы), которые являются частично палиндромными. Повторяющиеся последовательности (обычно специфические по отношению к виду) отделены вариабельными последовательностями постоянной длины (обычно от 20 до 58, в зависимости от CRISPR-локуса (WO 2007/024097, опубликованная 1 марта 2007 г.).
CRISPR-локусы впервые были идентифицированы у E. coli (Ishino et al. (1987) J. Bacterial. 169:5429-5433; Nakata et al. (1989) J. Bacterial. 171:3553-3556). Подобные рассеянные короткие повторяющиеся последовательности были идентифицированы у Haloferax mediterranei, Streptococcus pyogenes, Anabaena и Mycobacterium tuberculosis (Groenen et al. (1993) Mol. Microbiol. 10:1057-1065; Hoe et al. (1999) Emerg. Infect. Dis. 5:254-263; Masepohl et al. (1996) Biochim. Biophys. Acta 1307:26-30; Mojica et al. (1995) Mol. Microbiol. 17:85-93). CRISPR-локусы отличаются от других SSR структурой повторов, которые были названы короткими равномерно разделенными повторами (SRSR) (Janssen et al. (2002) OMICS J. Integ. Biol. 6:23-33; Mojica et al. (2000) Mol. Microbiol. 36:244-246). Повторы представляют собой короткие элементы, которые встречаются в кластерах, которые всегда равномерно разделены вариабельными последовательностями постоянной длины (Mojica et al. (2000) Mol. Microbiol. 36:244-246).
Термины “ген Cas”, “CRISPR-ассоциированный (Cas) ген” используются в данном документе взаимозаменяемо. Исчерпывающий обзор о семействе белков Cas представлен в Haft et al. (2005) Computational Biology, PLoS Comput Biol 1(6): e60. doi:10.1371/journal.pcbi.0010060. Как описано в данном документе, описано 41 семейство CRISPR-ассоциированных (Cas) генов, помимо четырех ранее известных семейств генов. Показано, что CRISPR-системы принадлежат к разным классам, с различными паттернами повторов, наборами генов и совокупностями видов. Число генов Cas в определенном CRISPR-локусе может различаться между видами.
Используемый в данном документе термин "Cas эндонуклеаза" относится к белку Cas, кодируемому геном Cas, причем указанный белок Cas способен к введению двунитевого разрыва в целевую последовательность ДНК. Эндонуклеаза Cas разматывает дуплекс ДНК вблизи геномного целевого сайта и расщепляет обе нити ДНК после распознавания целевой последовательности направляющей РНК, но только если правильный прилегающий к протоспейсеру мотив (PAM) примерно ориентирован на 3'-конце целевой последовательности.
Используемый в данном документе термин "направляющая РНК" относится к синтетическому слиянию двух молекул РНК, crRNA (РНК CRISPR), содержащей вариабельный направляющий домен, и tracrRNA. В одном варианте осуществления направляющая РНК содержит вариабельный направляющий домен из последовательностей с от 12 до 30 нуклеотидов и фрагмент РНК, который может взаимодействовать с эндонуклеазой Cas.
Выражение “вариабельный направляющий домен” относится к нуклеотидной последовательности 5-прим мотива последовательности GUUUU в направляющей РНК, которая является комплементарной одной цепи двунитевого целевого сайта ДНК в геноме растительной клетки, растения или семени. В одном варианте осуществления вариабельный направляющий домен имеет длину от 12 до 30 нуклеотидов.
В одном варианте осуществления направляющая РНК и эндонуклеаза Cas способны образовывать комплекс, который позволяет эндонуклеазе Cas вводить двунитевой разрыв в целевой сайт ДНК.
Используемый в данном документе термин "сайт распознавания" относится к последовательности ДНК, в которой индуцируется двунитевой разрыв в геноме клетки с помощью редкощепящего средства для индукции двунитевого разрыва. Термины "сайт распознавания", "последовательность распознавания" используются в данном документе взаимозаменяемо. Сайт распознавания может быть эндогенным сайтом в геноме хозяина (такого как дрожжи или растение), или в качестве альтернативы, сайт распознавания может быть гетерологичным по отношению к хозяину (дрожжам или растению), и, таким образом, не встречаться в природе в геноме, или сайт распознавания может находиться в гетерологичном местоположении в геноме по сравнению с тем, в котором он находится в природе.
Применяемый в данном документе термин "эндогенный сайт распознавания" относится к сайту распознавания редкощепящего средства для индукции двунитевого разрыва, который является эндогенным или нативным по отношению к геному хозяина (такого как растение или дрожжи) и расположен в эндогенном или нативном положении для такого сайта распознавания в геноме хозяина (такого как растение или дрожжи). Длина сайта распознавания может варьировать, при этом включены, например, сайты распознавания, имеющие длину, составляющую по меньшей мере 4, 6, 8, 10, 12, 14, 16, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70 или более нуклеотидов. Кроме того, возможно, что сайт распознавания может быть палиндромным, то есть последовательность одной нити читается точно так же в обратном направлении на комплементарной нити. Сайт однонитевого разрыва/расщепления может находиться в пределах последовательности распознавания, или сайт однонитевого разрыва/расщепления может находиться за пределами последовательности распознавания. В другом варианте расщепление может происходить в положениях нуклеотидов непосредственно напротив друг друга с получением разреза с тупыми концами, или, в других случаях, разрезы могут быть несимметрично расположенными с получением однонитевых выступающих концов, также называемых "липкими концами", которые могут быть 5'-липкими концами или 3'-липкими концами.
Используемый в данном документе термин "предполагаемый сайт распознавания" относится к последовательности распознавания, на которую было направлено сконструированное редкощепящее средство для индукции двунитевого разрыва, такое как сконструированная мегануклеаза, для специфического распознавания и индукции двунитевого разрыва. В одном варианте осуществления редкощепящее средство для индукции двунитевого разрыва представляет собой сконструированную мегануклеазу LIG3-4 (SEQ ID NO: 2), которая была разработана для распознавания предполагаемой последовательности распознавания SEQ ID NO: 13 (публикация заявки на патент США 2009-0133152 A1, опубликованной 21 мая 2009 года). В другом варианте осуществления редкощепящее средство для индукции двунитевого разрыва представляет собой сконструированную мегануклеазу MHP14+ (SEQ ID NO: 4), которая была разработана для распознавания предполагаемой последовательности распознавания SEQ ID NO: 14 (в заявке на патент США 13/427138, поданной 22 марта 2012 года).
Используемый в данном документе термин "вариантный сайт распознавания" относится к вариантной нуклеотидной последовательности, которая содержит изменение по меньшей мере по одному нуклеотидному основанию по сравнению с предполагаемым сайтом распознавания, на который было направлено сконструированное редкощепящее средство для индукции двунитевого разрыва, такое как мегануклеаза, для специфического распознавания и индукции двунитевого разрыва. Такое "изменение" включает, например: (i) замещение по меньшей мере одного нуклеотида, (ii) делецию по меньшей мере одного нуклеотида, (iii) вставку по меньшей мере одного нуклеотида или (iv) любую комбинацию из (i)-(iii). Активные варианты и фрагменты сайта распознавания могут характеризоваться по меньшей мере 65%, 70%, 75%, 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или большей идентичностью последовательности по отношению к данной последовательности распознавания, при этом активные варианты сохраняют биологическую активность и, следовательно, способны распознаваться и расщепляться эндонуклеазой. Вариантные сайты распознавания могут содержать по меньшей мере один (1) и вплоть до 2, 3, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 предпочтений к посторонним нуклеотидам. В одном варианте осуществления вариантные сайты распознавания являются неэндогенными по отношению к геному хозяина, такие вариантные сайты распознавания включают без ограничения вариантные сайты распознавания маиса, показанные на фигуре 4 (SEQ ID NO: 15-22 и SEQ ID NO: 23-35). В другом варианте осуществления вариантные сайты распознавания присутствуют в геноме хозяина (называются геномными вариантными сайтами распознавания) или являются эндогенными по отношению к геному хозяина, как например, геномы растений или дрожжей. В некоторых вариантах осуществления вариантные сайты распознавания можно ввести в геном растения с помощью мутагенеза эндогенной геномной последовательности. Способы сайт-специфического мутагенеза геномной ДНК известны из уровня техники и включают способы, описанные, например, в патенте США № 5565350, патенте США № 5731181 и патенте США № 6870075. Другие способы включают применение нуклеаз "цинковые пальцы", как например, те способы, описанные в публикации патентного документа США 20050208489.
"Геномный вариантный сайт распознавания" относится к вариантному сайту распознавания редкощепящего средства для индукции двунитевого разрыва, такого как мегануклеаза, который является эндогенным по отношению к геному организма (такого как растение или дрожжи). Одним примером вариантного сайта распознавания, который является эндогенным по отношению к геному маиса, является SEQ ID NO: 11.
Термины "предпочтительные посторонние нуклеотиды" или "предпочтения к посторонним нуклеотидам" можно использовать взаимозаменяемо, и они относятся к нуклеотидам, которые расположены в том же положении относительно нуклеотидов предполагаемого сайта распознавания, но которые являются более распространенными в идентифицированных геномных вариантных сайтах распознавания (см., например, распространенность для +8T (80%) в сравнении с предполагаемым сайтом распознавания +8C (13%) на фигуре 3A). В большинстве случаев предпочтительный посторонний нуклеотид, когда его помещают в предполагаемый сайт распознавания, расщепляется в большем проценте случаев, чем предполагаемый сайт распознавания (см., например, +8T (96% расщепление) в сравнении с +8C (80% расщепление на фигуре 5A).
В одном варианте осуществления предполагаемая последовательность распознавания сконструированной мегануклеазой LIG3-4 содержит SEQ ID NO: 13, тогда как вариантный сайт распознавания сконструированной мегануклеазой LIG3-4 содержит SEQ ID NO: 15, 16, 17, 18, 19, 20, 21 или 22. В другом варианте осуществления предполагаемая последовательность распознавания сконструированной мегануклеазой MHP14+ содержит SEQ ID NO: 14, тогда как вариантный сайт распознавания сконструированной мегануклеазой MHP14+ содержит SEQ ID NO: 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34 или 35.
"Локус вариантного сайта распознавания" представляет собой положение на хромосоме, содержащей вариабельный сайт распознавания. Предпочтительно, локус вариантного сайта распознавания находится в пределах 0, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 пар оснований от вариантного сайта распознавания.
Используемый в данном документе термин "мегануклеазная активность" относится к способности мегануклеазы производить двунитевой разрыв в желаемой последовательности распознавания и, таким образом, к сохранению активности в отношении индукции двунитевого разрыва. Получение двунитевого разрыва в последовательности распознавания или другой ДНК может называться в данном документе "разрезанием" или "расщеплением" последовательности распознавания или другой ДНК.
Известны анализы мегануклеазной активности, и в них, как правило, измеряют общую активность и специфичность мегануклеазы на ДНК-субстратах, содержащих сайты распознавания. Эти ДНК-субстраты включают без ограничения геномную ДНК и плазмидную ДНК. Например, мегануклеазную активность можно измерить in-vitro, как описано в данном документе в примере 3 и примере 9. Вкратце, процедуры расщепления в динамике можно выполнять при 37°C, 28°C и 23°C (или любой температуре, находящейся в диапазоне между 37°C, 36°C¸ 35°C¸ 34°C¸ 33°C¸ 32°C¸ 31°C¸ 30°C¸ 29°C¸ 28°C¸ 27°C¸ 26°C¸ 25°C¸ 24°C и 23°C) на плазмидной или геномной ДНК, содержащей сайт распознавания мегануклеазой, и % расщепления в каждом образце (также называемый % расщепления или % потери сайтов распознавания мегануклеазой) (являющийся показателем мегануклеазной активности) можно определить с помощью ПЦР в реальном времени.
Мегануклеазную активность также можно измерить с использованием скрининг-анализа на дрожжах, который описан в данном документе (фигуры 8 и 9 и пример 16). Вкратце, клетки дрожжей с функциональным геном Ade2 являются белыми, тогда как таковые, у которых отсутствует функционирование Ade2, проявляют красную окраску в связи с накоплением метаболита на более раннем этапе в пути биосинтеза аденина, что в результате приводит к образованию красных колоний с белыми секторами. Степень образования белых секторов, иногда распространяющегося на целые колонии, указывает на величину активности разрезания, присущей мегануклеазам. Поскольку фенотип образования секторов является качественным показателем мегануклеазной активности, задействовали числовую систему количественных показателей 0-4. Как показано на фигуре 3, количественный показатель 0 означает, что белые сектора не наблюдались (отсутствовало разрезание мегануклеазами); количественный показатель 4 означает полностью белые колонии (полное разрезание сайта распознавания); количественные показатели 1-3 означают промежуточные фенотипы образования белых секторов (и промежуточные степени разрезания сайта распознавания).
Дополнительно, мегануклеазную активность можно измерять in planta путем определения частоты мутаций в целевом сайте (TS). Частота мутаций в целевом сайте определяется следующим образом: (количество трансгенных объектов с модификацией в целевом сайте/общее количество трансгенных объектов)*100%.
Термины "повышенная" или "увеличенная" активность применяются в данном документе взаимозаменяемо. "Повышенная" или "увеличенная" мегануклеазная активность включает любое статистически значимое повышение активности исходного полипептида с мегануклеазной активностью, определяемое посредством любого из анализов активности, описанных в данном документе.
Мегануклеаза может быть предоставлена посредством полинуклеотида, кодирующего эндонуклеазу. Такой полинуклеотид, кодирующий эндонуклеазу, можно модифицировать для замены кодонов, имеющих более высокую частоту использования в растении по сравнению с встречающейся в природе последовательностью полинуклеотида. Например, полинуклеотид, кодирующий мегануклеазу, можно модифицировать для замены кодонов с более высокой частотой использования в растении маиса или сои по сравнению с встречающейся в природе последовательностью полинуклеотида.
Термины "контрольная мегануклеаза" или "эталонная мегануклеаза" можно использовать взаимозаменяемо, и они относятся к любой мегануклеазе, с которой сравнивают вариантную мегануклеазу. Контрольные мегануклеазы могут включать, без ограничений, исходные или соответствующие мегануклеазы или любые мегануклеазы дикого типа типа I-CreI.
Нумерация полимера на основе аминокислот или нуклеотидов, такого как любая из мегануклеаз по настоящему изобретению, соответствует нумерации выбранного полимера на основе аминокислот или нуклеиновой кислоты, если положение данного мономерного компонента (аминокислотного остатка, включенного нуклеотида и т.д.) полимера соответствует положению того же остатка в выбранном эталонном полипептиде или полинуклеотиде.
Как используется в данном документе, "участок генома, представляющий интерес" представляет собой сегмент хромосомы в геноме растения, введение полинуклеотида, представляющего интерес, или признака, представляющего интерес, в который является желательным. Участок генома, представляющий интерес, может включать, например, один или несколько полинуклеотидов, представляющих интерес. Как правило, участок генома, представляющий интерес, по настоящему изобретению содержит сегмент хромосомы, размер которого составляет 0-15 сантиморганид (cM).
Используемый в данном документе термин "полинуклеотид, представляющий интерес", в участке генома, представляющем интерес, представляет собой любую кодирующую и/или некодирующую часть участка генома, представляющего интерес, в том числе без ограничения трансгена, нативного гена, мутированного гена и генетического маркера, такого как, например, маркер однонуклеотидного полиморфизма (SNP) и маркер простой повторяющейся последовательности (SSR).
Как используется в данном документе, "физически сцепленный", "в физическом сцеплении" и "генетически сцепленный" используются для обозначения любых двух или более генов, трансгенов, нативных генов, мутированных генов, изменений, целевых сайтов, маркеров и подобного, которые являются частью одной молекулы ДНК или хромосомы.
Как применяется в данном документе, "выделенный" полинуклеотид или полипептид или его биологически активная часть являются по существу или практически свободными от компонентов, обычно сопутствующих полинуклеотиду или полипептиду или взаимодействующих с ними, которые обнаруживаются в его природной среде. Таким образом, выделенный или очищенный полинуклеотид или полипептид является по существу свободным от другого клеточного материала или культуральной среды при получении с помощью методик рекомбинантных молекул или по существу свободными от химических предшественников или других химических веществ при химическом синтезе. В оптимальном случае "выделенный" полинуклеотид не содержит последовательностей (в оптимальном случае последовательностей, кодирующих белок), которые в естественных условиях фланкируют полинуклеотид (т.е. последовательностей, расположенных на 5'- и 3'-концах полинуклеотида) в геномной ДНК организма, из которого получен полинуклеотид. Например, в различных вариантах осуществления выделенный полинуклеотид может содержать нуклеотидную последовательность размером менее чем приблизительно 5 т.о., 4 т.о., 3 т.о., 2 т.о., 1 т.о., 0,5 т.о. или 0,1 т.о., которая в естественных условиях фланкирует полинуклеотид в геномной ДНК клетки, из которой получен полинуклеотид. Полипептид, который по существу свободен от клеточного материала, включает препараты полипептидов, имеющие менее чем примерно 30%, 20%, 10%, 5% или 1% (по сухому весу) загрязняющего белка. Если полипептид по настоящему изобретению или его биологически активная часть получены рекомбинантным путем, то, в оптимальном случае, в культуральной среде представлено менее чем примерно 30%, 20%, 10%, 5% или 1% (по сухому весу) химических предшественников или химических веществ, не являющихся белком, представляющим интерес.
Как применяется в данном документе, полинуклеотид или полипептид являются "рекомбинантными", если они являются искусственными или сконструированными или получены из искусственных или сконструированных белка или нуклеиновой кислоты. Например, полинуклеотид, вставляемый в вектор или любое другое гетерологичное местоположение, например, в геном рекомбинантного организма, так, что он не связан с нуклеотидными последовательностями, которые в естественных условиях фланкируют полинуклеотид, как это обнаруживается в природе, является рекомбинантным полинуклеотидом. Полипептид, образовавшийся в результате in vivo или in vitro экспрессии рекомбинантного полинуклеотида, является примером рекомбинантного полипептида. Аналогично, последовательность полинуклеотида, которая не встречается в природе, например, вариант встречающегося в природе гена, является рекомбинантной.
"Подпоследовательность" или "фрагмент" означает любую часть полной последовательности.
Сравнения последовательностей
Для описания взаимоотношений последовательностей двух или более полинуклеотидов или полипептидов используют следующие термины: (a) "эталонная последовательность", (b) "окно сравнения", (c) "идентичность последовательностей" и (d) "процентная идентичность последовательностей".
(a) Как применяется в данном документе, "эталонная последовательность" является заданной последовательностью, применяемой в качестве основы для сравнения последовательностей. Эталонная последовательность может быть частью определенной последовательности или всей таковой; например, в виде сегмента кДНК полной длины или последовательности гена или целой кДНК, или последовательности гена, или белковой последовательности.
(b) Как применяется в данном документе, "окно сравнения" относится к смежному и определенному сегменту последовательности полипептида, где последовательность полипептида в окне сравнения может включать добавления или делеции (т.е. гэпы) по сравнению с эталонной последовательностью (которая не содержит добавления или делеции) для оптимального выравнивания двух полипептидов. Обычно длина окна сравнения составляет по меньшей мере 5, 10, 15 или 20 смежных аминокислот, или она может составлять 30, 40, 50, 100 или более. Специалисты в данной области понимают, что во избежание высокой степени подобия по отношению к эталонной последовательности вследствие включения гэпов в последовательность полипептида, как правило, вводится штраф за открытие гэпа, и его вычитают из количества совпадений.
Способы выравнивания последовательностей для сравнения хорошо известны в уровне техники. Таким образом, определение процентной идентичности последовательности между любыми двумя последовательностями можно выполнить с использованием математического алгоритма. Неограничивающими примерами таких математических алгоритмов являются алгоритм по Myers and Miller (1988) CABIOS 4:11-17; алгоритм локального выравнивания по Smith et al. (1981) Adv. Appl. Math. 2:482; алгоритм глобального выравнивания по Needleman and Wunsch (1970) J. Mol. Biol. 48:443-453; способ поиска локального выравнивания по Pearson and Lipman (1988) Proc. Natl. Acad. Sci. 85:2444-2448; алгоритм по Karlin and Altschul (1990) Proc. Natl. Acad. Sci. USA 872264; модифицированный по Karlin and Altschul (1993) Proc. Natl. Acad. Sci. USA 90:5873-5877. Для сравнения последовательностей можно использовать компьютерные реализации данных математических алгоритмов, чтобы определить идентичность последовательностей. Такие реализации включают без ограничений CLUSTAL в программе PC/Gene (доступна от Intelligenetics, Маунтин-Вью, Калифорния); программу ALIGN (версия 2.0) и GAP, BESTFIT, BLAST, FASTA и TFASTA в GCG Wisconsin Genetics Software Package, версия 10 (доступны от Accelrys Inc., 9685 Scranton Road, Сан-Диего, Калифорния, США). Выравнивания при помощи этих программ могут быть выполнены с использованием параметров по умолчанию. Программа CLUSTAL хорошо описана в Higgins et al. (1988) Gene 73:237-244 (1988); Higgins et al. (1989) CABIOS 5:151-153; Corpet et al. (1988) Nucleic Acids Res. 16:10881-90; Huang et al. (1992) CABIOS 8:155-65; и Pearson et al. (1994) Meth. Mol. Biol. 24:307-331. Программа ALIGN основана на алгоритме из Myers and Miller (1988), упоминаемом выше. При сравнении аминокислотных последовательностей в программе ALIGN можно применять таблицу весов замен остатков PAM120, штраф за продолжение гэпа 12 и штраф за открытие гэпа 4. Программы BLAST по Altschul et al (1990) J. Mol. Biol. 215:403; основаны на алгоритме Karlin and Altschul (1990), упоминаемом выше. Поиски нуклеотидных последовательностей в BLAST можно выполнять с помощью программы BLASTN при параметрах вес выравнивания = 100, длина слова = 12 с получением нуклеотидных последовательностей, гомологичных нуклеотидной последовательности, кодирующей белок согласно настоящему изобретению. Поиски белковых последовательностей в BLAST можно выполнять с помощью программы BLASTX при параметрах вес выравнивания = 50, длина слова = 3 с получением аминокислотных последовательностей, гомологичных белку или полипептиду согласно настоящему изобретению. Поиски белковых последовательностей в BLASTP можно выполнять с использованием параметров по умолчанию. См. blast.ncbi.nlm.nih.gov/Blast.cgi.
Выравнивания последовательностей и расчеты процентного подобия можно производить с помощью программы Megalign из комплекта биоинформационных вычислительных программ LASARGENE (DNASTAR Inc., Мэдисон, Висконсин) или с помощью программы AlignX из комплекта биоинформационных вычислительных программ Vector NTI (Invitrogen, Карлсбад, Калифорния). Множественное выравнивание последовательностей выполняют с использованием способа выравнивания Clustal (Higgins and Sharp, CABIOS. 5:151-153 (1989)) с параметрами по умолчанию (ШТРАФ ЗА ОТКРЫТИЕ ГЭПА=10, ШТРАФ ЗА ПРОДОЛЖЕНИЕ ГЭПА=10). Параметры по умолчанию для парного выравнивания и расчета процента идентичности белковых последовательностей с помощью программы Clustal являются следующими: РАЗМЕР ИДЕНТИЧНОГО УЧАСТКА=1, ШТРАФ НА ВНЕСЕНИЕ ДЕЛЕЦИИ=3, ДЛИНА СЕГМЕНТА=5 и ЧИСЛО НЕПРЕРЫВНО СОВПАДАЮЩИХ УЧАСТКОВ=5. Для нуклеиновых кислот эти параметры являются следующими: ШТРАФ ЗА ОТКРЫТИЕ ГЭПА=10, ШТРАФ ЗА ПРОДОЛЖЕНИЕ ГЭПА=10, KTUPLE=2, ШТРАФ ЗА ОТКРЫТИЕ ГЭПА=5, ОКНО=4 и СОХРАНЕННЫЕ ДИАГОНАЛИ=4. "Значительная часть" аминокислотной или нуклеотидной последовательности включает достаточную часть аминокислотной последовательности полипептида или нуклеотидной последовательности гена для обеспечения предполагаемой идентификации этого полипептида или гена путем оценки последовательности специалистом в данной области техники вручную или путем автоматизированного при помощи компьютера сравнения и идентификации последовательностей с помощью алгоритмов, таких как BLAST (Altschul, S. F. et al., J. Mol. Biol. 215:403-410 (1993)) и BLAST с гэпами (Altschul, S. F. et al., Nucleic Acids Res. 25:3389-3402 (1997)). BLASTN относится к программе BLAST, сравнивающей запрашиваемую нуклеотидную последовательность с нуклеотидными последовательностями из базы данных.
"Ген" относится к фрагменту нуклеиновой кислоты, который экспрессирует специфический белок, включая регуляторные последовательности, предшествующие (5'-некодирующие последовательности) и идущие после (3'-некодирующие последовательности) кодирующей последовательности. Термин "нативный ген" означает ген, как находится в природе, со своими собственными регуляторными последовательностями. "Химерный ген" или "рекомбинантная экспрессионная конструкция", применяемые взаимозаменяемо, относятся к любому гену, который не является нативным геном, включающему регуляторные и кодирующие последовательности, которые вместе не встречаются в природе. Соответственно, химерный ген может содержать регуляторные последовательности и кодирующие последовательности, которые получены из различных источников, или регуляторные последовательности и кодирующие последовательности, которые получены из одного и того же источника, но расположены в порядке, отличающемся от встречающегося в природе. "Эндогенный ген" относится к нативному гену в его естественном расположении в геноме организма. "Чужеродный" ген относится к гену, не обнаруживаемому в норме в организме хозяина, но который введен в организм хозяина с помощью переноса гена. Чужеродные гены могут включать нативные гены, введенные в организм, не являющийся нативным, или химерные гены. "Трансген" является геном, который ввели в геном с помощью способа трансформации.
"Кодирующая последовательность" относится к последовательности ДНК, которая кодирует конкретную аминокислотную последовательность. "Регуляторные последовательности" относятся к нуклеотидным последовательностям, расположенным выше (5'-некодирующие последовательности), в пределах или ниже (3'-некодирующие последовательности) кодирующей последовательности, и влияющим на транскрипцию, процессинг или стабильность РНК или трансляцию связанной кодирующей последовательности. Регуляторные последовательности могут включать без ограничения промоторы, лидерные последовательности трансляции, интроны и последовательности распознавания полиаденилирования.
"Вырожденность кодонов" относится к отклонению в генетическом коде, позволяющему производить изменение нуклеотидной последовательности без влияния на аминокислотную последовательность кодируемого полипептида. Соответственно, настоящее изобретение относится к любому фрагменту нуклеиновой кислоты, содержащему нуклеотидную последовательность, которая кодирует все аминокислотные последовательности, изложенные в данном документе, или их значительную часть. Специалисту в данной области техники хорошо известно о "смещении частоты использования кодонов", проявляемом конкретной клеткой-хозяином в отношении использования нуклеотидных кодонов, определяющих данную аминокислоту. Таким образом, при синтезе фрагмента нуклеиновой кислоты для повышенной экспрессии в клетке-хозяине желательно разработать такой фрагмент нуклеиновой кислоты, чтобы его частота использования кодонов была близка к частоте использования предпочтительных кодонов клетки-хозяина.
Как используется в данном документе, "идентичность последовательности" или "идентичность" применительно к двум последовательностям полинуклеотидов или полипептидов относится к остаткам двух последовательностей, которые являются одинаковыми при выравнивании с нахождением максимального соответствия в указанном окне сравнения. Если процент идентичности последовательностей применяют в отношении белков, то учитывают, что положения остатков, которые не являются идентичными, часто отличаются по консервативным аминокислотным заменам, где аминокислотные остатки заменены на другие аминокислотные остатки со сходными химическими свойствами (например, зарядом или гидрофобностью). Если последовательности отличаются консервативными заменами, то процент идентичности последовательности можно повысить с тем, чтобы внести поправку на консервативную природу замены. Говорят, что последовательности, которые отличаются такими консервативными заменами, характеризуются "сходством последовательностей" или "сходством". Средства для осуществления такой корректировки хорошо известны специалистам в данной области. Как правило, это предусматривает оценку в баллах консервативной замены скорее в качестве частичного, чем полного несовпадения, что, таким образом, увеличивает процент идентичности последовательностей. Таким образом, например, если идентичной аминокислоте присваивается балл 1, а неконсервативной замене присваивается балл ноль, то консервативной замене присваивается балл от ноля до 1. Оценку в баллах консервативных замен рассчитывают, например, как реализовано в программе PC/GENE (Intelligenetics, Маунтин-Вью, Калифорния).
Как применяется в данном документе, "процент идентичности последовательностей" означает значение, определяемое путем сравнения двух выровненных последовательностей в окне сравнения, где часть последовательности полинуклеотида в окне сравнения может включать добавления или делеции (т.е., гэпы) по сравнению с эталонной последовательностью (которая не включает добавления или делеции) для оптимального выравнивания двух последовательностей. Процентное значение рассчитывают путем определения количества положений, в которых идентичный остаток нуклеиновой кислоты или аминокислотный остаток встречается у обеих последовательностей, с получением количества совпавших положений, деления количества совпавших положений на общее количество положений в окне сравнения и умножения результата на 100 с получением процентной идентичности последовательностей.
Полинуклеотидные конструкции
В данном документе предусматриваются молекулы полинуклеотидов или нуклеиновых кислот, содержащие вариантные сайты распознавания для редкощепящих средств для индукции двунитевого разрыва или любых их активных вариантов или фрагментов. Термины "полинуклеотид", "последовательность полинуклеотида", "последовательность нуклеиновой кислоты" и "фрагмент нуклеиновой кислоты" применяются в данном документе взаимозаменяемо. Эти выражения охватывают нуклеотидные последовательности и т.п. Использование термина "полинуклеотид" не подразумевает ограничение настоящего изобретения полинуклеотидами, содержащими ДНК. Специалисты в данной области поймут, что полинуклеотиды могут содержать рибонуклеотиды и комбинации рибонуклеотидов и дезоксирибонуклеотидов. Такие дезоксирибонуклеотиды и рибонуклеотиды включают в себя как встречающиеся в природе молекулы, так и синтетические аналоги. Полинуклеотиды по настоящему изобретению также охватывают все формы последовательностей, в том числе без ограничений однонитевые формы, двунитевые формы, шпильки, структуры типа "стебель-петля" и т.п.
Кроме того, предусматриваются рекомбинантные полинуклеотиды, содержащие различные редкощепящие средства для индукции двунитевого разрыва, такие как сконструированные мегануклеазы. Термины "рекомбинантный полинуклеотид", "рекомбинантный нуклеотид", "рекомбинантная ДНК" и "рекомбинантная ДНК-конструкция" используются в данном документе взаимозаменяемо. Рекомбинантная конструкция содержит комбинацию искусственных или гетерологичных последовательностей нуклеиновых кислот, например, регуляторных и кодирующих последовательностей, которые не встречаются вместе в природе. Например, кассета для переноса может содержать сайты рестрикции и гетерологичный полинуклеотид, представляющий интерес. В других вариантах осуществления рекомбинантная конструкция может содержать регуляторные последовательности и кодирующие последовательности, которые получены из разных источников, или регуляторные последовательности и кодирующие последовательности, которые получены из одного и того же источника, но расположены в порядке, отличающемся от встречающегося в природе. Такую конструкцию можно применять сам по себе или можно применять в сочетании с вектором. Если используют вектор, тогда выбор вектора зависит от способа, который будут использовать для трансформации клеток-хозяев, что хорошо известно специалистам в данной области. Например, можно применять плазмидный вектор. Специалисту в данной области хорошо известны генетические элементы, которые должны присутствовать в векторе для успешной трансформации, отбора и размножения клеток-хозяев, содержащих какой-либо из выделенных фрагментов нуклеиновых кислот, обеспечиваемых в данном документе. Специалисту в данной области также будет понятно, что у различных независимых трансформантов будут проявляться различные уровни и паттерны экспрессии (Jones et al., EMBO J. 4:2411-2418 (1985); De Almeida et al., Mol. Gen. Genetics 218:78-86 (1989)), и таким образом, для того, чтобы получить линии, проявляющие желаемые уровень и паттерн экспрессии, следует провести скрининг нескольких трансгенных объектов. Такой скрининг, помимо прочего, можно выполнять с помощью Саузерн-анализа ДНК, Нозерн-анализа экспрессии мРНК, анализа экспрессии белка с помощью иммуноблоттинга или фенотипического анализа.
Полинуклеотиды, кодирующие мегануклеазы, раскрытые в данном документе, могут быть представлены в кассетах экспрессии для экспрессии в растении, представляющем интерес. Кассета может включать в себя 5'- и 3'-регуляторные последовательности, функционально связанные с полинуклеотидом, кодирующим мегануклеазу или ее активный вариант или фрагмент. Подразумевается, что термин "функционально связанный" означает функциональную связь между двумя или более элементами. Например, функциональная связь между полинуклеотидом, представляющим интерес, и регуляторной последовательностью (т.е. промотором) представляет собой функциональную связь, которая обеспечивает экспрессию полинуклеотида, представляющего интерес. Функционально связанные элементы могут быть смежными или несмежными. При использовании для обозначения соединения двух участков, кодирующих белок, под функционально связанным подразумевается, что кодирующие участки находятся в одной и той же рамке считывания. Кассета может дополнительно содержать по меньшей мере один дополнительный ген, подлежащий введению в организм путем котрансформации. Альтернативно, дополнительный (дополнительные) ген(ы) могут быть представлены в нескольких кассетах экспрессии. Такая кассета экспрессии обеспечивается множеством сайтов рестрикции и/или сайтов рекомбинации для того, чтобы вставка полинуклеотида, кодирующего мегануклеазу или ее активный вариант или фрагмент, подвергалась регуляции транскрипции со стороны регуляторных областей. Кассета экспрессии может дополнительно содержать гены селектируемых маркеров.
В направлении транскрипции 5'-3' кассета экспрессии может содержать участок инициации транскрипции и трансляции (т.е. промотор), полинуклеотид, кодирующий мегануклеазу или ее активный вариант или фрагмент, и участок терминации транскрипции и трансляции (т.е. участок терминации), являющиеся функциональными у растений. Регуляторные участки (т.е. промоторы, участки регуляции транскрипции и участки терминации трансляции) и/или полинуклеотид, кодирующий мегануклеазу или ее активный вариант или фрагмент, могут быть нативными/аналогичными по отношению к клетке-хозяину или друг к другу. Альтернативно, регуляторные участки и/или полинуклеотид, кодирующий мегануклеазу или ее активный вариант или фрагмент, могут быть гетерологичными по отношению к клетке-хозяину или друг к другу.
Используемая в данном документе "гетерологичная" по отношению к последовательности означает, что последовательность происходит из чужеродного вида или, если она происходит из того же вида, в существенной степени модифицирована по составу и/или местоположению в геноме по сравнению с ее нативной формой в результате преднамеренного вмешательства человека. Например, промотор, функционально связанный с гетерологичным полинуклеотидом, происходит из вида, отличного от вида, из которого получен полинуклеотид, или, если он происходит из того же/аналогичного вида, то один или оба из них являются в значительной степени модифицированными по сравнению с их исходной формой и/или местоположением в геноме, или промотор не является нативным промотором для функционально связанного полинуклеотида.
Несмотря на то, что экспрессия последовательностей с помощью гетерологичных промоторов может быть оптимальной, можно применять нативные промоторные последовательности. Такие конструкции могут изменять уровни экспрессии полинуклеотида, кодирующего мегануклеазу, в растении или растительной клетке. Таким образом, фенотип растения или растительной клетки можно изменять.
Участок терминации может быть нативным по отношению к участку инициации транскрипции, может быть нативным по отношению к функционально связанному полинуклеотиду, кодирующему мегануклеазу или ее активный вариант или фрагмент, может быть нативным по отношению к растению-хозяину или может быть получен из другого источника (т.е. чужеродного или гетерологичного по отношению к промотору, полинуклеотиду, кодирующему мегануклеазу или ее активный вариант или фрагмент, растению-хозяину или любой их комбинации). Подходящие участки терминации доступны из Ti-плазмиды A. tumefaciens, например, участка терминации генов октопинсинтазы и нопалинсинтазы. См. также Guerineau et al. (1991) Mol. Gen. Genet. 262:141-144; Proudfoot (1991) Cell 64:671-674; Sanfacon et al. (1991) Genes Dev. 5:141-149; Mogen et al. (1990) Plant Cell 2:1261-1272; Munroe et al. (1990) Gene 91:151-158; Ballas et al. (1989) Nucleic Acids Res. 17:7891-7903; и Joshi et al. (1987) Nucleic Acids Res. 15:9627-9639.
При необходимости полинуклеотиды можно оптимизировать для повышения экспрессии в трансформированном растении. То есть полинуклеотиды можно синтезировать с использованием кодонов, предпочтительных для растения, для улучшения экспрессии. См., например, Campbell and Gowri (1990) Plant Physiol. 92:1-11; в отношении рассмотрения использования кодонов, предпочтительных для хозяина. Из уровня техники доступны способы синтеза генов, предпочтительных для растений. См., например, патенты США №№ 5380831 и 5436391; и Murray et al. (1989) Nucleic Acids Res. 17:477-498, включенной в данный документ посредством ссылки.
Известны дополнительные модификации последовательности для усиления экспрессии гена у клеточного хозяина. Они включают исключение последовательностей, кодирующих ложные сигналы полиаденилирования, сигналы сайта сплайсинга экзонов и интронов, транспозоноподобные повторы и другие подобные хорошо изученные последовательности, которые могут оказывать пагубное влияние на экспрессию гена. Содержание G-C в последовательности можно корректировать до уровней, средних для данного клеточного хозяина, что рассчитывается с учетом известных генов, экспрессируемых в клетке-хозяине. Если возможно, последовательность модифицируют для того, чтобы избежать образования прогнозируемых шпилечных вторичных структур мРНК.
Кассеты экспрессии могут дополнительно содержать 5'-лидерные последовательности. Такие лидерные последовательности могут способствовать усилению трансляции. Трансляционные лидерные последовательности известны в данной области и включают лидерные последовательности пикорнавирусов, например, лидерную последовательность EMCV (5'-некодирующий участок генома вируса энцефаломиокардита) (Elroy-Stein et al. (1989) Proc. Natl. Acad. Sci. USA 86:6126-6130); лидерные последовательности потивирусов, например, лидерную последовательность TEV (вируса гравировки табака) (Gallie et al. (1995) Gene 165(2):233-238), лидерную последовательность MDMV (вируса карликовой мозаики кукурузы) (Virology 154:9-20) и мРНК белка, связывающего тяжелую цепь иммуноглобулина человека (BiP) (Macejak et al. (1991) Nature 353:90-94); нетранслируемую лидерную последовательность мРНК белка оболочки вируса мозаики люцерны (РНК-4 AMV) (Jobling et al. (1987) Nature 325:622-625); лидерную последовательность вируса табачной мозаики (TMV) (Gallie et al. (1989) в Molecular Biology of RNA, ed. Cech (Liss, New York), pp. 237-256) и лидерную последовательность вируса хлоротической пятнистости маиса (MCMV) (Lommel et al. (1991) Virology 81:382-385. См. также Della-Cioppa et al. (1987) Plant Physiol. 84:965-968.
При получении кассеты экспрессии с различными фрагментами ДНК можно производить манипуляции так, чтобы получить последовательности ДНК в надлежащей ориентации и, при необходимости, в надлежащей рамке считывания. С этой целью для соединения фрагментов ДНК можно использовать адаптеры или линкеры, или можно задействовать другие манипуляции для обеспечения подходящих сайтов рестрикции, удаления избыточной ДНК, удаления сайтов рестрикции и т.п. С этой целью можно задействовать мутагенез in vitro, репарацию с помощью праймеров, рестрикцию, отжиг, повторные замены, например, транзиции и трансверсии.
Для экспрессии различных последовательностей мегануклеаз, раскрытых в данном документе, можно применять ряд промоторов, в том числе нативный промотор последовательности полинуклеотида, представляющей интерес. Промоторы можно выбирать, исходя из требуемого результата. Такие промоторы включают, например, конститутивные, предпочтительные для тканей или другие промоторы для экспрессии в растениях.
Конститутивные промоторы включают, например, коровый промотор промотора Rsyn7 и другие конститутивные промоторы, раскрытые в WO 99/43838 и патенте США № 6072050; коровый промотор 35S CaMV (Odell et al. (1985) Nature 313:810-812); актиновый промотор риса (McElroy et al. (1990) Plant Cell 2:163-171); убиквитиновый промотор (Christensen et al. (1989) Plant Mol. Biol. 12:619-632; и Christensen et al. (1992) Plant Mol. Biol. 18:675-689); pEMU (Last et al. (1991) Theor. Appl. Genet. 81:581-588); MAS (Velten et al. (1984) EMBO J. 3:2723-2730); промотор ALS (патент США № 5659026) и т.п. Другие конститутивные промоторы включают, например, описанные в патентах США №№ 5608149; 5608144; 5604121; 5569597; 5466785; 5399680; 5268463; 5608142 и 6177611.
Для нацеливания на повышенную экспрессию мегануклеаз в конкретной ткани растения можно использовать предпочтительные для тканей промоторы. Предпочтительные для ткани промоторы включают описанные у Yamamoto et al. (1997) Plant J. 12(2):255-265; Kawamata et al. (1997) Plant Cell Physiol. 38(7):792-803; Hansen et al. (1997) Mol. Gen Genet. 254(3):337-343; Russell et al. (1997) Transgenic Res. 6(2):157-168; Rinehart et al. (1996) Plant Physiol. 112(3):1331-1341; Van Camp et al. (1996) Plant Physiol. 112(2):525-535; Canevascini et al. (1996) Plant Physiol. 112(2):513-524; Yamamoto et al. (1994) Plant Cell Physiol. 35(5):773-778; Lam (1994) Results Probl. Cell Differ. 20:181-196; Orozco et al. (1993) Plant Mol Biol. 23(6):1129-1138; Matsuoka et al. (1993) Proc Natl. Acad. Sci. USA 90(20):9586-9590; и Guevara-Garcia et al. (1993) Plant J. 4(3):495-505. В случае необходимости, такие промоторы можно модифицировать с получением слабой экспрессии.
Промоторы, активные преимущественно в листьях, известны из уровня техники. См., например, Yamamoto et al. (1997) Plant J. 12(2):255-265; Kwon et al. (1994) Plant Physiol. 105:357-67; Yamamoto et al. (1994) Plant Cell Physiol. 35(5):773-778; Gotor et al. (1993) Plant J. 3:509-18; Orozco et al. (1993) Plant Mol. Biol. 23(6):1129-1138; и Matsuoka et al. (1993) Proc. Natl. Acad. Sci. USA 90(20):9586-9590.
Для экспрессии последовательностей, кодирующих мегануклеазы, или их биологически активных вариантов и фрагментов можно применять синтетические промоторы.
Кассета экспрессии может также содержать ген селектируемого маркера для отбора трансформированных клеток. Гены селектируемых маркеров используются для отбора трансформированных клеток или тканей. Маркерные гены включают гены, кодирующие устойчивость к антибиотикам, как, например, таковые, кодирующие неомицинфосфотрансферазу II (NEO) и гигромицинфосфотрансферазу (HPT), а также гены, придающие устойчивость к гербицидным соединениям, таким как глифосат, глюфосинат аммония, бромоксинил, сульфонилмочевины, дикамба и 2,4-дихлорфеноксиацетат (2,4-D). Дополнительные селектируемые маркеры включают фенотипические маркеры, такие как β-галактозидаза и флуоресцентные белки, такие как зеленый флуоресцентный белок (GFP) (Su et al. (2004) Biotechnol Bioeng 85:610-9; и Fetter et al. (2004) Plant Cell 16:215-28), голубой флуоресцентный белок (CYP) (Bolte et al. (2004) J. Cell Science 117:943-54; и Kato et al. (2002) Plant Physiol 129:913-42) и желтый флуоресцентный белок (PhiYFP™ от Evrogen, см. Bolte et al. (2004) J. Cell Science 117:943-54). О дополнительных селектируемых маркерах см., в целом, в Yarranton (1992) Curr. Opin. Biotech. 3:506-511; Christopherson et al. (1992) Proc. Natl. Acad. Sci. USA 89:6314-6318; Yao et al. (1992) Cell 71:63-72; Reznikoff (1992) Mol. Microbiol. 6:2419-2422; Barkley et al. (1980) в The Operon, pp. 177-220; Hu et al. (1987) Cell 48:555-566; Brown et al. (1987) Cell 49:603-612; Figge et al. (1988) Cell 52:713-722; Deuschle et al. (1989) Proc. Natl. Acad. Aci. USA 86:5400-5404; Fuerst et al. (1989) Proc. Natl. Acad. Sci. USA 86:2549-2553; Deuschle et al. (1990) Science 248:480-483; Gossen (1993) Ph.D. Thesis, University of Heidelberg; Reines et al. (1993) Proc. Natl. Acad. Sci. USA 90:1917-1921; Labow et al. (1990) Mol. Cell. Biol. 10:3343-3356; Zambretti et al. (1992) Proc. Natl. Acad. Sci. USA 89:3952-3956; Baim et al. (1991) Proc. Natl. Acad. Sci. USA 88:5072-5076; Wyborski et al. (1991) Nucleic Acids Res. 19:4647-4653; Hillenand-Wissman (1989) Topics Mol. Struc. Biol. 10:143-162; Degenkolb et al. (1991) Antimicrob. Agents Chemother. 35:1591-1595; Kleinschnidt et al. (1988) Biochemistry 27:1094-1104; Bonin (1993) Ph.D. Thesis, University of Heidelberg; Gossen et al. (1992) Proc. Natl. Acad. Sci. USA 89:5547-5551; Oliva et al. (1992) Antimicrob. Agents Chemother. 36:913-919; Hlavka et al. (1985) Handbook of Experimental Pharmacology, Vol. 78 (Springer-Verlag, Berlin); Gill et al. (1988) Nature 334:721-724. Такие раскрытия включены в данный документ посредством ссылки. Приведенный выше перечень генов селектируемых маркеров не предназначен для ограничения. В настоящем изобретении можно применять любой ген селектируемого маркера.
Способ введения
Редкощепящее средство для индукции двунитевого разрыва, такое как мегануклеаза, можно вводить с помощью любых средств, известных из уровня техники. Например, предусматриваются клетка, дрожжи или растение, имеющие предполагаемый или вариантный сайт распознавания в своем геноме. Можно обеспечивать транзиентную экспрессию мегануклеазы, или полипептид сам по себе можно непосредственно доставлять в клетку. Альтернативно, нуклеотидная последовательность, способная к экспрессии мегануклеазы, может быть стабильно интегрирована в геном растения. При наличии соответствующего предполагаемого или вариантного сайта распознавания и мегануклеазы донорную ДНК можно вводить в геном трансформированного растения. Альтернативно, различные компоненты можно объединять путем полового скрещивания трансформированных растений. Таким образом, последовательность, кодирующую мегануклеазу, и/или предполагаемый или вариантный сайт распознавания можно объединять друг с другом в ходе полового скрещивания с обеспечением присутствия каждого компонента системы в одном растении. Мегануклеаза может находиться под контролем конститутивного или индуцируемого промотора. Такие промоторы, представляющие интерес, обсуждаются более подробно в другом месте в данном документе.
Для введения последовательности, представляющей интерес, такой как любое из редкощепящих средств для индукции двунитевого разрыва, в растение или часть растения можно применять различные способы. Подразумевается, что "введение" означает предоставление растению, растительной клетке или части растения полинуклеотида или полипептида таким образом, что последовательность попадает внутрь клетки растения. Способы по настоящему изобретению не зависят от конкретного способа введения последовательности в растение или часть растения, за исключением того, что полинуклеотид или полипептиды попадают внутрь по меньшей мере одной клетки растения. Из уровня техники известны способы введения полинуклеотида и/или полипептидов в растения, в том числе без ограничений способы стабильной трансформации, способы транзиентной трансформации и способы трансформации, опосредованной вирусами.
Подразумевается, что "стабильная трансформация" означает, что нуклеотидный конструкция, вводимая в растение, интегрируется в геном растения и может быть унаследована его потомками. Подразумевается, что "транзиентная трансформация" означает, что полинуклеотид вводится в растение и не интегрируется в геном растения или в растение вводится полипептид.
Протоколы трансформации, а также протоколы введения полипептидов или последовательностей полинуклеотидов в растения могут меняться в зависимости от типа растения или растительной клетки, т.е. однодольного или двудольного растения, которое целенаправленно трансформируют. Подходящие способы введения полипептидов и полинуклеотидов в растительные клетки включают микроинъекцию (Crossway et al. (1986) Biotechniques 4:320-334), электропорацию (Riggs et al. (1986) Proc. Natl. Acad. Sci. USA 83:5602-5606, Agrobacterium-опосредованную трансформацию (патент США № 5563055 и патент США № 5981840), прямой перенос генов (Paszkowski et al. (1984) EMBO J. 3:2717-2722) и баллистическое ускорение частиц (см., например, патент США № 4945050; патент США № 5879918; патент США № 5886244 и № 5932782; Tomes et al. (1995) в Plant Cell, Tissue and Organ Culture: Fundamental Methods, ed. Gamborg and Phillips (Springer-Verlag, Berlin); McCabe et al. (1988) Biotechnology 6:923-926); а также трансформацию при помощи Lec1 (WO 00/28058). Также см. Weissinger et al. (1988) Ann. Rev. Genet. 22:421-477; Sanford et al. (1987) Particulate Science and Technology 5:27-37 (лук); Christou et al. (1988) Plant Physiol. 87:671-674 (соя); McCabe et al. (1988) Bio/Technology 6:923-926 (soybean); Finer and McMullen (1991) In Vitro Cell Dev. Biol. 27P:175-182 (соя); Singh et al. (1998) Theor. Appl. Genet. 96:319-324 (соя); Datta et al. (1990) Biotechnology 8:736-740 (рис); Klein et al. (1988) Proc. Natl. Acad. Sci. USA 85:4305-4309 (маис); Klein et al. (1988) Biotechnology 6:559-563 (маис); патенты США №№ 5240855, 5322783 и 5324646; Klein et al. (1988) Plant Physiol. 91:440-444 (маис); Fromm et al. (1990) Biotechnology 8:833-839 (маис); Hooykaas-Van Slogteren et al. (1984) Nature (London) 311:763-764; патент США № 5736369 (злаки); Bytebier et al. (1987) Proc. Natl. Acad. Sci. USA 84:5345-5349 (Liliaceae); De Wet et al. (1985) в The Experimental Manipulation of Ovule Tissues, ed. Chapman et al. (Longman, New York), pp. 197-209 (пыльца); Kaeppler et al. (1990) Plant Cell Reports 9:415-418 и Kaeppler et al. (1992) Theor. Appl. Genet. 84:560-566 (опосредованная нитевидными кристаллами трансформация); D'Halluin et al. (1992) Plant Cell 4:1495-1505 (электропорация); Li et al. (1993) Plant Cell Reports 12:250-255; и Christou and Ford (1995) Annals of Botany 75:407-413 (рис); Osjoda et al. (1996) Nature Biotechnology 14:745-750 (маис, при помощи Agrobacterium tumefaciens); все из которых включены в данный документ посредством ссылки.
В конкретных вариантах осуществления последовательность редкощепящего средства для индукции двунитевого разрыва, такую как последовательность мегануклеазы, или ее активный вариант или фрагменты можно доставлять в дрожжевую клетку или растение при помощи различных способов транзиентной трансформации. Такие способы транзиентной трансформации включают, без ограничений, введение белка мегануклеазы или его активных вариантов и фрагментов непосредственно в дрожжевую клетку или растение. Такие способы включают, например, микроинъекцию или бомбардировку частицами. См., например, Crossway et al. (1986) Mol Gen. Genet. 202:179-185; Nomura et al. (1986) Plant Sci. 44:53-58; Hepler et al. (1994) Proc. Natl. Acad. Sci. 91: 2176-2180; и Hush et al. (1994) The Journal of Cell Science 107:775-784, все из которых включены в данный документ посредством ссылки.
Как правило, такие способы включают встраивание нуклеотидной конструкции по настоящему изобретению в молекулу ДНК или РНК. Понятно, что последовательность мегануклеазы можно изначально синтезировать как часть вирусного полипротеина, который затем можно подвергать процессингу путем протеолиза in vivo или in vitro с получением требуемого рекомбинантного белка. Кроме того, подразумевается, что промоторы по настоящему изобретению также охватывают промоторы, используемые для транскрипции вирусными РНК-полимеразами. Способы введения в растения полинуклеотидов, в том числе молекул вирусной ДНК или РНК, и способы экспрессии белка, кодируемого ими, известны из уровня техники. См., например, патенты США №№ 5889191, 5889190, 5866785, 5589367, 5316931; и Porta et al. (1996) Molecular Biotechnology 5:209-221; включенные в данный документ посредством ссылки.
Из уровня техники известны способы целенаправленной вставки полинуклеотида в конкретное местоположение в геноме растения. В одном варианте осуществления вставку полинуклеотида в требуемое местоположение в геноме выполняют с использованием системы сайт-специфичной рекомбинации. См., например, WO 99/25821, WO 99/25854, WO 99/25840, WO 99/25855 и WO 99/25853, все из которых включены в данный документ посредством ссылки. Вкратце, полинуклеотид по настоящему изобретению может содержаться в кассете для переноса, фланкированной двумя нерекомбиногенными сайтами рекомбинации. Кассету для переноса вводят в растение, имеющее в своем геноме стабильно встроенный целевой сайт, фланкированный двумя нерекомбиногенными сайтами рекомбинации, которые соответствуют сайтам кассеты для переноса. Обеспечивают подходящую рекомбиназу, и кассета для переноса интегрируется в целевой сайт. Полинуклеотид, представляющий интерес, таким образом, интегрируется в конкретное хромосомное положение в геноме растения. Другие способы нацеливания полинуклеотидов изложены в WO 2009/114321 (включенной в данный документ посредством ссылки), в котором описаны "оригинальные" мегануклеазы, получаемые для модификации геномов растений, в частности, генома маиса. См. также Gao et al. (2010) Plant Journal 1:176-187.
Из клеток, которые были трансформированы, можно вырастить растения в соответствии с традиционными способами. См., например, McCormick et al. (1986) Plant Cell Reports 5:81-84. Эти растения можно затем выращивать и опылять с помощью либо той же самой трансформированной линии, либо других линий, и идентифицировать полученных в результате потомков, которые характеризуются конститутивным проявлением указанной требуемой фенотипической характеристики. Можно вырастить два или более поколений, чтобы убедиться в том, что экспрессия требуемой фенотипической характеристики стабильно поддерживается и наследуется, а затем собрать семена, чтобы убедиться в том, что была достигнута экспрессия требуемой фенотипической характеристики. Таким образом, в настоящем изобретении предусмотрено трансформированное семя (также называемое "трансгенным семенем"), имеющее полинуклеотид по настоящему изобретению, например, кассету экспрессии по настоящему изобретению, стабильно встроенную в его геном.
Как используется в данном документе, "праймеры" представляют собой выделенные полинуклеотиды, гибридизирующиеся с комплементарной нитью целевой ДНК путем гибридизации нуклеиновых кислот с образованием гибрида праймера и целевой нити ДНК, которые затем удлиняют вдоль нити целевой ДНК с помощью полимеразы, например, ДНК-полимеразы. Пары праймеров по настоящему изобретению относят к их использованию в амплификации целевого полинуклеотида, например, посредством полимеразной цепной реакции (ПЦР) или других традиционных способов амплификации нуклеиновых кислот. "ПЦР" или "полимеразная цепная реакция" представляет собой методику, применяемую для амплификации конкретных сегментов ДНК (см. патенты США №№ 4683195 и 4800159; включенные в данный документ посредством ссылки).
Зонды и праймеры имеют достаточную длину в нуклеотидах, чтобы связываться с целевой последовательностью ДНК и обеспечивать специфическое выявление и/или идентификацию полинуклеотида, кодирующего полипептид с мегануклеазной активностью или его активный вариант или фрагмент, описанные в другом месте в данном документе. Понятно, что условия гибридизации или условия реакции для достижения данного результата могут определяться оператором. Эта длина может быть любой длиной, которая является достаточной длиной для того, чтобы быть применимой в предпочтительном способе выявления. Такие зонды и праймеры могут осуществлять специфическую гибридизацию с целевой последовательностью в условиях гибридизации высокой жесткости. Зонды и праймеры согласно вариантам осуществления настоящего изобретения могут обладать полной идентичностью смежных нуклеотидов в последовательности ДНК по отношению к целевой последовательности, хотя с помощью традиционных способов можно разработать зонды, отличающиеся от целевой последовательности ДНК и сохраняющие способность к обеспечению специфического выявления и/или идентификации целевой последовательности ДНК. Соответственно, зонды и праймеры могут обладать примерно 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или большей идентичностью последовательностей или комплементарностью по отношению к целевому полинуклеотиду.
Способы получения и применения зондов и праймеров описаны, например, в Molecular Cloning: A Laboratory Manual, 2.sup.nd ed, vol. 1-3, ed. Sambrook et al., Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y. 1989 (далее, "Sambrook et al., 1989"); Current Protocols in Molecular Biology, ed. Ausubel et al., Greene Publishing and Wiley-Interscience, New York, 1992 (с периодическими обновлениями) (далее, "Ausubel et al., 1992"); и Innis et al., PCR Protocols: A Guide to Methods and Applications, Academic Press: San Diego, 1990. Пары праймеров для ПЦР можно получать из известной последовательности, например, путем применения компьютерных программ, предназначенных для этой цели, таких как средство анализа праймеров для ПЦР в Vector NTI версии 10 (Invitrogen); PrimerSelect (DNASTAR Inc., Мэдисон, Висконсин) и Primer (версия 0.5.COPYRGT., 1991, институт биомедицинских исследований Уайтхеда, Кэмбридж, Массачусетс). Дополнительно, последовательности можно подвергать зрительному сканированию, а праймеры идентифицировать вручную с применением руководства, известного специалисту в данной области.
Дрожжи и растения
Предусматриваются дрожжи, растения, растительные клетки, части и семена растений, а также зерно, имеющие вариантные последовательности распознавания для редкощепящих средств для индукции двунитевого разрыва, таких как мегануклеазы, которые раскрыты в данном документе. В конкретных вариантах осуществления дрожжи, растения и/или части растений имеют стабильно встроенный по меньшей мере одну гетерологичную вариантную последовательность распознавания, раскрытую в данном документе, или ее активный вариант или фрагмент. Таким образом, обеспечиваются дрожжи, растения, растительные клетки, части и семена растений, содержащие по меньшей мере одну вариантную последовательность распознавания с любым из SEQ ID NO: 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35 или любой их комбинацией, или их биологически активный фрагмент и/или вариант. В конкретных вариантах осуществления вариантные последовательности распознавания проявляют повышенную активность расщепления в отношении редкощепящего средства для индукции двунитевого разрыва.
Используемое в данном документе выражение "растение" включает растительные клетки, протопласты растительных клеток, тканевые культуры растительных клеток, из которых можно регенерировать растения, растительные каллюсы, скопления растительных клеток и растительные клетки, которые являются интактными в растениях или частях растений, таких как зародыши, пыльца, семяпочки, семена, листья, цветки, ветви, плоды, зерна, колоски, початки, листовые обвертки, стебли, корни, кончики корней, пыльники и т.п. Предполагается, что зерно означает зрелое семя, полученное коммерческими растениеводами для целей, отличных от выращивания или воспроизводства вида. Потомки, варианты и мутанты регенерированных растений также включены в объем настоящего изобретения, при условии, что эти части содержат введенные полинуклеотиды.
Трансформированное растение или трансформированная растительная клетка, обеспечиваемые в данном документе, представляют собой таковые, в которых генетическое изменение, такое как трансформация, затронуло ген, представляющий интерес, или представляют собой растение или растительную клетку, которые происходят от растения или клетки, измененных таким образом, и которые содержат изменение. "Трансген" является геном, который ввели в геном с помощью способа трансформации. В соответствии с этим, "трансгенное растение" представляет собой растение, которое содержит трансген, независимо от того, ввели ли трансген в данное конкретное растение путем трансформации или скрещивания; таким образом, потомки первоначально трансформированного растения охвачены определением. “Контроль”, или “контрольное растение”, или “контрольная растительная клетка” обеспечивают точку отсчета для измерения изменений фенотипа исследуемого растения или растительной клетки. Контрольное растение или растительная клетка могут включать, например, (a) растение или клетку дикого типа, т.е., с тем же генотипом, что и у исходного материала для генетического изменения, из которого в результате получили исследуемое растение или клетку; (b) растение или растительную клетку с тем же генотипом, что и у исходного материала, но которые трансформировали с помощью нулевой конструкции (т.е. с помощью конструкции, которая не экспрессирует трансген, такой как конструкция, содержащая маркерный ген); (c) растение или растительную клетку, которые представляют собой нетрансформированные сегреганты среди потомков исследуемых растения или растительной клетки; (d) растение или растительную клетку, генетически идентичные исследуемым растению или растительной клетке, но которые не подвергаются воздействию условий или раздражителей, которые будут индуцировать экспрессию трансгена; или (e) исследуемые растение или растительную клетку как таковые в условиях, в которых конструкция не экспрессируется.
Из растительных клеток, которые трансформировали для экспрессии мегануклеазы, обеспечиваемой в данной документе, можно вырастить целые растения. Регенерация, развитие и культивирование растений из трансформированных протопластов из одного растения или из различных трансформированных эксплантов хорошо известны в данном уровне техники. См., например, McCormick et al. (1986) Plant Cell Reports 5:81-84; Weissbach and Weissbach, в Methods for Plant Molecular Biology, (Eds.), Academic Press, Inc. San Diego, Calif., (1988). Данный способ регенерации и выращивания, как правило, включает этапы отбора трансформированных клеток, культивирования этих индивидуализированных клеток на протяжении обычных стадий развития зародыша до стадии укоренившегося саженца. Трансгенные зародыши и семена регенерируют подобным образом. Полученные в результате укоренившиеся трансгенные побеги впоследствии высаживают в соответствующую среду для выращивания растений, такую как почва. Предпочтительно, чтобы регенерированные растения были самоопыляющимися для получения гомозиготных трансгенных растений. В других случаях пыльцой, полученной из регенерированных растений, перекрестно опыляют выращенные из семян растения агрономически важных линий. В свою очередь, пыльцу от растений данных важных линий используют для опыления регенерированных растений. Можно вырастить два или более поколений, чтобы убедиться в том, что экспрессия требуемой фенотипической характеристики стабильно поддерживается и наследуется, а затем собрать семена, чтобы убедиться в том, что была достигнута экспрессия требуемой фенотипической характеристики. Таким образом, композиции, представленные в данном документе, обеспечивают трансформированное семя (также называемое “трансгенным семенем”), имеющее полинуклеотид, обеспечиваемый в данном документе, например, целевой сайт, стабильно встроенный в его геном.
Вариантные последовательности распознавания и их активные варианты и фрагменты, раскрытые в данном документе, можно использовать для трансформации любых видов растений, в том числе, без ограничений, однодольных и двудольных. Примеры видов растений, представляющих интерес, включают без ограничений кукурузу (Zea mays), Brassica sp. (например, B. napus, B. rapa, B. juncea), в частности, те виды Brassica, которые являются пригодными в качестве источников растительного масла, люцерну (Medicago sativa), рис (Oryza sativa), рожь (Secale cereale), сорго (Sorghum bicolor, Sorghum vulgare), просо (например, пеннисетум рогозовидный (Pennisetum glaucum), просо культурное (Panicum miliaceum), щетинник итальянский (Setaria italica), просо пальчатое (Eleusine coracana)), подсолнечник (Helianthus annuus), сафлор (Carthamus tinctorius), пшеницу (Triticum aestivum), сою (Glycine max), табак (Nicotiana tabacum), картофель (Solanum tuberosum), разновидности арахиса (Arachis hypogaea), хлопчатник (Gossypium barbadense, Gossypium hirsutum), сладкий картофель (Ipomoea batatus), маниок (Manihot esculenta), кофейное дерево (Coffea spp.), кокосовую пальму (Cocos nucifera), ананас (Ananas comosus), цитрусовые деревья (Citrus spp.), шоколадное дерево (Theobroma cacao), чайный куст (Camellia sinensis), банан (Musa spp.), авокадо (Persea americana), инжир (Ficus casica), гуаву (Psidium guajava), манго (Mangifera indica), маслину (Olea europaea), папайю (Carica papaya), кешью (Anacardium occidentale), макадамию (Macadamia integrifolia), миндаль (Prunus amygdalus), разновидности сахарной свеклы (Beta vulgaris), сахарный тростник (Saccharum spp.), разновидности овса, ячмень, овощи, декоративные растения и хвойные растения.
Овощи включают разновидности томата (Lycopersicon esculentum), латук (например, Lactuca sativa), разновидности зеленой фасоли (Phaseolus vulgaris), разновидности лимской фасоли (Phaseolus limensis), разновидности гороха (Lathyrus spp.) и представителей рода Cucumis, таких как огурец (C. sativus), канталупа (C. cantalupensis) и дыня мускусная (C. melo). Декоративные растения включают азалию (Rhododendron spp.), гортензию (Macrophylla hydrangea), гибискус (Hibiscus rosasanensis), розы (Rosa spp.), тюльпаны (Tulipa spp.), нарциссы (Narcissus spp.), петунии (Petunia hybrida), гвоздику (Dianthus caryophyllus), пуансеттию (Euphorbia pulcherrima) и хризантему.
Хвойные растения, которые можно использовать при практическом осуществлении настоящего изобретения, включают, например, виды сосны, такие как сосна ладанная (Pinus taeda), сосна Эллиота (Pinus elliotii), сосна желтая (Pinus ponderosa), сосна скрученная широкохвойная (Pinus contorta) и сосна лучистая (Pinus radiata); псевдотсугу Мензиса (Pseudotsuga menziesii); тсугу канадскую (Tsuga canadensis); ель белую (Picea glauca); секвойю вечнозеленую (Sequoia sempervirens); истинные пихты, такие как пихта миловидная (Abies amabilis) и пихта бальзамическая (Abies balsamea), и туи, такие как туя гигантская (Thuja plicata) и каллитропсис нутканский (Chamaecyparis nootkatensis), а также тополь и эвкалипт. В конкретных вариантах осуществления растения по настоящему изобретению являются культурными растениями (например, кукуруза, люцерна, подсолнечник, Brassica, соя, хлопчатник, сафлор, арахис, сорго, пшеница, просо, табак и т.д.). В других вариантах осуществления растения кукурузы и сои являются оптимальными, а еще в нескольких других вариантах осуществления оптимальными являются растения кукурузы.
Другие растения, представляющие интерес, включают зерновые растения, которые обеспечивают семена, представляющие интерес, масличные растения и бобовые растения. Семена, представляющие интерес, включают семена зерновых культур, таких как кукуруза, пшеница, ячмень, рис, сорго, рожь и т.д. Масличные растения включают хлопчатник, сою, сафлор, подсолнечник, Brassica, маис, люцерну, пальму, кокосовую пальму и т.д. Бобовые растения включают бобы и разновидности гороха. Бобы включают гуар, рожковое дерево, пажитник, сою, разновидности обыкновенной фасоли, вигну китайскую, золотистую фасоль, лимскую фасоль, стручковую фасоль, разновидности чечевицы, турецкий горох и т.д.
Неограничивающие примеры композиций и способов, раскрытых в данном документе, являются следующими.
1) Способ идентификации вариантного сайта распознавания для редкощепящего сконструированного средства для индукции двунитевого разрыва, способного к введению редкого двунитевого разрыва в предполагаемом сайте распознавания, причем указанный способ включает:
a) приведение в контакт геномной ДНК с редкощепящим сконструированным средством для индукции двунитевого разрыва, способным к введению двунитевого разрыва в указанную геномную ДНК, где двунитевой разрыв приводит в результате к образованию нуклеотидного липкого конца;
b) лигирование первого адаптера с указанным нуклеотидным липким концом;
c) разрезание лигированной ДНК, полученной на стадии (b), и лигирование по меньшей мере одного второго адаптера с разрезанным нуклеотидным концом для обеспечения амплификации и секвенирования фрагментов геномной ДНК, окружающих двунитевой разрыв;
d) выравнивание нуклеотидных последовательностей фрагментов ДНК, полученных в (c), с эталонной геномной последовательностью ДНК; и
e) идентификацию вариантного сайта распознавания, содержащего изменение по меньшей мере по одному нуклеотидному основанию по сравнению с предполагаемым сайтом распознавания указанного сконструированного средства для индукции двунитевого разрыва.
2) Способ согласно варианту осуществления 1, где редкощепящее сконструированное средство для индукции двунитевого разрыва выбирают из группы, состоящей из мегануклеазы, нуклеазы "цинковые пальцы", TAL-эффекторной нуклеазы, транспозазы и сайт-специфической рекомбиназы.
3) Способ согласно варианту осуществления 1, где нуклеотидный липкий конец представляет собой 3'-нуклеотидный липкий конец.
4) Способ согласно варианту осуществления 1, где нуклеотидный липкий конец представляет собой 5'-нуклеотидный липкий конец.
5) Способ согласно варианту осуществления 1, где первый адаптер, лигированный с нуклеотидным липким концом, представляет собой нефосфорилированный по 5' адаптер.
6) Способ согласно варианту осуществления 1, где геномную ДНК выбирают из группы, состоящей из прокариотической ДНК, эукариотической ДНК и синтетической ДНК.
7) Способ согласно варианту осуществления 6, где эукариотическую ДНК выделяют из растения, дрожжей или животного.
8) Способ согласно варианту осуществления 7, где растение выбирают из группы, состоящей из сои, подсолнечника, хлопчатника, люцерны, канолы, хлопчатника, табака, картофеля, Arabidopsis, сафлора, маиса, риса, сорго, ячменя, пшеницы, проса, овса, сахарного тростника, газонной травы и проса прутьевидного.
9) Способ согласно варианту осуществления 1, где средство для индукции двунитевого разрыва получают из I-CreI.
10) Способ идентификации вариантного сайта распознавания с улучшенной активностью расщепления для редкощепящего сконструированного средства для индукции двунитевого разрыва, способного к введению редкого двунитевого разрыва в предполагаемом сайте распознавания, причем указанный способ включает:
a) приведение в контакт геномной ДНК с редкощепящим сконструированным средством для индукции двунитевого разрыва, способным к введению двунитевого разрыва в указанную геномную ДНК, где двунитевой разрыв приводит в результате к образованию нуклеотидного липкого конца;
b) лигирование первого адаптера с указанным нуклеотидным липким концом;
c) разрезание лигированной ДНК, полученной на стадии (b), и лигирование второго адаптера с разрезанным нуклеотидным концом для обеспечения амплификации и секвенирования фрагментов геномной ДНК, окружающих двунитевой разрыв;
d) выравнивание нуклеотидных последовательностей фрагментов ДНК, полученных в (c), с эталонной геномной последовательностью ДНК; и
e) идентификацию вариантного сайта распознавания, содержащего изменение по меньшей мере по одному нуклеотидному основанию по сравнению с предполагаемым сайтом распознавания указанного редкощепящего сконструированного средства для индукции двунитевого разрыва;
f) анализ активности редкощепящего сконструированного средства для индукции двунитевого разрыва в вариантных сайтах распознавания из d);
g) идентификацию вариантного сайта распознавания, который приводит в результате к повышенной активности редкощепящего сконструированного средства для индукции двунитевого разрыва по сравнению с активностью в предполагаемом сайте распознавания.
11) Способ согласно варианту осуществления 10, где о повышенной активности редкощепящего сконструированного средства для индукции двунитевого разрыва свидетельствует:
a) более высокий процент (%) расщепления вариантного сайта распознавания по сравнению с процентом (%) расщепления предполагаемого сайта распознавания, где сайты распознавания расположены на геномной ДНК;
b) более высокий процент (%) расщепления вариантного сайта распознавания по сравнению с процентом (%) расщепления предполагаемого сайта распознавания, где сайты распознавания расположены на плазмидной ДНК;
c) более высокая оценка в анализе на дрожжах для вариантного сайта распознавания по сравнению с предполагаемым сайтом распознавания; или
d) любая комбинация (a), (b) и (c).
12) Способ введения в геном клетки вариантного сайта распознавания для редкощепящего сконструированного средства для индукции двунитевого разрыва, способного вводить редкий двунитевой разрыв в предполагаемый сайт распознавания, причем указанный способ включает:
a) обеспечение наличия донорной ДНК, содержащей вариантный сайт распознавания для редкощепящего сконструированного средства для индукции двунитевого разрыва, способного к введению двунитевого разрыва в предполагаемый сайт распознавания, где указанное редкощепящее сконструированное средство для индукции двунитевого разрыва также способно к введению двунитевого разрыва в указанный вариантный сайт распознавания;
b) обеспечение наличия растительной клетки;
c) приведение в контакт растительной клетки с донорной ДНК; и
d) идентификацию по меньшей мере одной растительной клетки из (c), содержащей в своем геноме указанный вариантный сайт распознавания.
13) Способ согласно варианту осуществления 12, где редкощепящее сконструированное средство для индукции двунитевого разрыва выбирают из группы, состоящей из мегануклеазы, нуклеазы "цинковые пальцы", TAL-эффекторной нуклеазы, транспозазы, эндонуклеазы Cas и сайт-специфической рекомбиназы.
14) Выделенный полинуклеотид, содержащий вариантный сайт распознавания с улучшенной активностью расщепления для сконструированной мегануклеазы, способной к введению двунитевого разрыва в предполагаемый сайт распознавания, где указанный вариантный сайт распознавания содержит нуклеотидную последовательность с заменой по меньшей мере 1 нуклеотидного основания по сравнению с предполагаемым сайтом распознавания SEQ ID NO: 14.
15) Выделенный полинуклеотид согласно варианту осуществления 1, где указанный вариантный сайт распознавания содержит последовательность с изменениями по меньшей мере 2, 3, 4, 5, 6 или 7 пар оснований по сравнению с SEQ ID NO: 14.
16) Выделенный полинуклеотид согласно варианту осуществления 14, где указанный вариантный сайт распознавания содержит:
a) аденин (A) в положении, соответствующем нуклеотидному положению 9 в SEQ ID NO: 14;
b) гуанин (G) в положении, соответствующем нуклеотидному положению 10 в SEQ ID NO: 14;
c) тимин (T) в положении, соответствующем нуклеотидному положению 11 в SEQ ID NO: 14;
d) аденин (A) в положении, соответствующем нуклеотидному положению 13 в SEQ ID NO: 14;
e) тимин (T) в положении, соответствующем нуклеотидному положению18 в SEQ ID NO: 14;
f) гуанин (G) в положении, соответствующем нуклеотидному положению 19 в SEQ ID NO: 14;
g) гуанин (G) или аденин (A) в положении, соответствующем нуклеотидному положению 22 в SEQ ID NO: 14; или
h) любую комбинацию из a)-g).
17) Выделенный полинуклеотид согласно варианту осуществления 14, где указанная вариантная последовательность распознавания выбрана из группы, состоящей из SEQ ID NO: 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34 и 35.
18) Выделенный полинуклеотид согласно варианту осуществления 14, где об улучшенной активности расщепления свидетельствует:
a) более высокий процент (%) расщепления вариантного сайта распознавания по сравнению с процентом (%) расщепления предполагаемого сайта распознавания SEQ ID NO:14, где сайты распознавания расположены на геномной ДНК;
b) более высокий процент (%) расщепления вариантного сайта распознавания по сравнению с процентом (%) расщепления предполагаемого сайта распознавания, где сайты распознавания расположены на плазмидной ДНК;
c) более высокая оценка в анализе на дрожжах для вариантного сайта распознавания по сравнению с предполагаемым сайтом распознавания; или
d) любая комбинация (a), (b) и (c).
19) Рекомбинантный фрагмент ДНК, содержащий выделенный полинуклеотид согласно варианту осуществления 14.
20) Клетка, содержащая рекомбинантный фрагмент ДНК согласно варианту осуществления 19.
21) Клетка согласно варианту осуществления 20, где клетка представляет собой клетку дрожжей или растительную клетку.
22) Трансгенное растение или семя, содержащее растительную клетку согласно варианту осуществления 21.
23) Трансгенное растение согласно варианту осуществления 22, где указанное растение выбрано из группы, состоящей из маиса, пшеницы, риса, ячменя, сахарного тростника, сорго, ржи, проса прутьевидного, сои, Brassica, подсолнечника, хлопчатника или люцерны.
24) Выделенный полинуклеотид, содержащий вариантный сайт распознавания с улучшенной активностью расщепления для сконструированной мегануклеазы, способной к введению двунитевого разрыва в предполагаемый сайт распознавания, где указанный вариантный сайт распознавания содержит нуклеотидную последовательность с заменой по меньшей мере 1 нуклеотидного основания по сравнению с предполагаемым сайтом распознавания SEQ ID NO: 13.
25) Выделенный полинуклеотид согласно варианту осуществления 24, где указанный вариантный сайт распознавания содержит последовательность с изменениями по меньшей мере 2, 3, 4 или 5 пар оснований по сравнению с SEQ ID NO: 13.
26) Выделенный полинуклеотид согласно варианту осуществления 24, где указанный вариантный сайт распознавания содержит:
a) цитозин (C) в положении, соответствующем нуклеотидному положению 1 в SEQ ID NO: 13
b) цитозин (C) в положении, соответствующем нуклеотидному положению 5 в SEQ ID NO: 13;
c) гуанин (G) в положении, соответствующем нуклеотидному положению 10 в SEQ ID NO: 13;
d) тимин (T) аденин (A) в положении, соответствующем нуклеотидному положению 11 в SEQ ID NO: 13;
e) тимин (T) в положении, соответствующем нуклеотидному положению 19 в SEQ ID NO: 13;
f) любую комбинацию из a)-e).
27) Выделенный полинуклеотид согласно варианту осуществления 24, где указанная вариантная последовательность распознавания выбрана из группы, состоящей из SEQ ID NO: 15, 16, 17, 18, 19, 20 и 21.
28) Выделенный полинуклеотид согласно варианту осуществления 24, где об улучшенной активности расщепления свидетельствует:
a) более высокий процент (%) расщепления вариантного сайта распознавания по сравнению с процентом (%) расщепления предполагаемого сайта распознавания SEQ ID NO:13, где сайты распознавания расположены на геномной ДНК;
b) более высокий процент (%) расщепления вариантного сайта распознавания по сравнению с процентом (%) расщепления предполагаемого сайта распознавания, где сайты распознавания расположены на плазмидной ДНК;
c) более высокая оценка в анализе на дрожжах для вариантного сайта распознавания по сравнению с предполагаемым сайтом распознавания; или
d) любая комбинация (a), (b) и (c).
29) Рекомбинантный фрагмент ДНК, содержащий выделенный полинуклеотид согласно варианту осуществления 24.
30) Клетка, содержащая рекомбинантный фрагмент ДНК согласно варианту осуществления 29.
31) Клетка согласно варианту осуществления 30, где клетка представляет собой клетку дрожжей или растительную клетку.
32) Трансгенное растение или семя, содержащее растительную клетку согласно варианту осуществления 31.
33) Трансгенное растение согласно варианту осуществления 32, где указанное растение выбрано из группы, состоящей из маиса, пшеницы, риса, ячменя, сахарного тростника, сорго, ржи, проса прутьевидного, сои, Brassica, подсолнечника, хлопчатника или люцерны.
34) Способ нацеливания вставки полинуклеотида, представляющего интерес, в специфический хромосомный сайт в геноме растения, причем указанный способ включает:
a) трансформацию растительной клетки или растения фрагментом ДНК, содержащим полинуклеотид, представляющий интерес, где указанный геном указанных растительной клетки или растения содержит по меньшей мере один вариантный сайт распознавания, выбранный из группы, состоящей из SEQ ID NO: 15, 16, 17, 18, 19, 20 и 21; и
b) обеспечение наличия мегануклеазы, способной производить двунитевой разрыв в вариабельном сайте распознавания из (a); и
c) отбор указанных растительной клетки или растения, содержащих указанный полинуклеотид, представляющий интерес, интегрированный в указанный вариантный сайт распознавания.
35) Способ согласно варианту осуществления 34, где обеспечение наличия указанной мегануклеазы включает интеграцию в геном указанных растительной клетки или растения нуклеотидной последовательности, кодирующей мегануклеазу SEQ ID NO: 1.
36) Растение или растительная клетка, полученные с помощью способа согласно варианту осуществления 34.
37) Способ нацеливания вставки полинуклеотида, представляющего интерес, в специфический хромосомный сайт в геноме растения, причем указанный способ включает:
a) трансформацию растительной клетки или растения фрагментом ДНК, содержащим полинуклеотид, представляющий интерес, где указанный геном указанных растительной клетки или растения содержит по меньшей мере один вариантный сайт распознавания, выбранный из группы, состоящей из SEQ ID NO: 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34 и 35;
b) обеспечение наличия мегануклеазы, способной производить двунитевой разрыв в вариабельном сайте распознавания из (a); и
c) отбор указанных растительной клетки или растения, содержащих указанный полинуклеотид, представляющий интерес, интегрированный в указанный вариантный сайт распознавания.
38) Способ согласно варианту осуществления 37, где обеспечение наличия указанной мегануклеазы включает интеграцию в геном указанных растительных клеток нуклеотидной последовательности, кодирующей мегануклеазу SEQ ID NO: 3
39) Растение или растительная клетка, полученные с помощью способа согласно варианту осуществления 37.
40) Способ идентификации вариантного сайта распознавания для редкощепящего сконструированного средства для индукции двунитевого разрыва, способного к введению редкого двунитевого разрыва в предполагаемом сайте распознавания, причем указанный способ включает:
a) приведение в контакт геномной ДНК с редкощепящим сконструированным средством для индукции двунитевого разрыва, способного к введению двунитевого разрыва в указанную геномную ДНК, где двунитевой разрыв приводит в результате к образованию тупого конца;
b) создание нуклеотидного липкого конца из тупого конца из (a);
c) лигирование первого адаптера с нуклеотидным липким концом из (b);
d) разрезание лигированной ДНК, полученной на стадии (c), и лигирование по меньшей мере одного второго адаптера с разрезанным нуклеотидным концом для обеспечения амплификации и секвенирования фрагментов геномной ДНК, окружающих двунитевой разрыв;
e) выравнивание нуклеотидных последовательностей фрагментов ДНК, полученных в (d), с эталонной геномной последовательностью ДНК; и
f) идентификацию вариантного сайта распознавания, содержащего изменение по меньшей мере по одному нуклеотидному основанию по сравнению с предполагаемым сайтом распознавания указанного сконструированного средства для индукции двунитевого разрыва.
41) Способ согласно варианту осуществления 40, где редкощепящее сконструированное средство для индукции двунитевого разрыва представляет собой эндонуклеазу Cas.
42) Способ согласно варианту осуществления 41, где эндонуклеаза Cas способна к образованию комплекса с направляющей РНК, где указанный комплекс позволяет эндонуклеазе Cas вводить двунитевой разрыв в указанную геномную ДНК.
43) Способ согласно варианту осуществления 40, где геномную ДНК выбирают из группы, состоящей из прокариотической ДНК, эукариотической ДНК и синтетической ДНК.
44) Способ согласно варианту осуществления 43, где эукариотическую ДНК выделяют из растения, дрожжей или животного.
45) Способ идентификации вариантного сайта распознавания с улучшенной активностью расщепления для редкощепящего сконструированного средства для индукции двунитевого разрыва, способного к введению редкого двунитевого разрыва в предполагаемом сайте распознавания, причем указанный способ включает:
a) приведение в контакт геномной ДНК с редкощепящим сконструированным средством для индукции двунитевого разрыва, способного к введению двунитевого разрыва в указанную геномную ДНК, где двунитевой разрыв приводит в результате к образованию тупого конца;
b) создание нуклеотидного липкого конца из тупого конца из (a);
c) лигирование первого адаптера с нуклеотидным липким концом из (b);
d) разрезание лигированной ДНК, полученной на стадии (c), и лигирование по меньшей мере одного второго адаптера с разрезанным нуклеотидным концом для обеспечения амплификации и секвенирования фрагментов геномной ДНК, окружающих двунитевой разрыв;
e) выравнивание нуклеотидных последовательностей фрагментов ДНК, полученных в (d), с эталонной геномной последовательностью ДНК; и
f) идентификацию вариантного сайта распознавания, содержащего изменение по меньшей мере по одному нуклеотидному основанию по сравнению с предполагаемым сайтом распознавания указанного редкощепящего сконструированного средства для индукции двунитевого разрыва;
g) анализ активности редкощепящего сконструированного средства для индукции двунитевого разрыва в вариантных сайтах распознавания из f); и
h) идентификацию вариантного сайта распознавания, который приводит в результате к повышенной активности редкощепящего сконструированного средства для индукции двунитевого разрыва по сравнению с активностью в предполагаемом сайте распознавания.
46) Способ согласно варианту осуществления 45, где редкощепящее сконструированное средство для индукции двунитевого разрыва представляет собой эндонуклеазу Cas.
47) Способ согласно варианту осуществления 46, где эндонуклеаза Cas способна к образованию комплекса с направляющей РНК, где указанный комплекс позволяет эндонуклеазе Cas вводить двунитевой разрыв в указанную геномную ДНК.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
ПРИМЕР 1
Создание редкощепящих сконструированных мегануклеаз
A. Мегануклеаза LIG3-4 и предполагаемая последовательность распознавания для LIG3-4
Эндогенный геномный целевой сайт маиса, содержащий предполагаемую последовательность распознавания для LIG3-4 (SEQ ID NO: 13), выбирали для разработки редкощепящего средства для индукции двунитевого разрыва (SEQ ID NO: 1), как описано в публикации заявки на патент США 2009-0133152 A1 (опубликованной 21 мая 2009 года). Предполагаемая последовательность распознавания для LIG3-4 представляет собой полинуклеотид размером 22 п.о., имеющий следующую последовательность: ATATACCTCACACGTACGCGTA (SEQ ID NO: 13).
B. Мегануклеазы MHP14+ и сайт распознавания для MHP14
Эндогенный геномный целевой сайт маиса, содержащий предполагаемую последовательность распознавания для MHP14+ (SEQ ID NO: 14), выбирали для разработки редкощепящего средства для индукции двунитевого разрыва (SEQ ID NO: 3), как описано в заявке на патент США 13/427138, поданной 22 марта 2012 года). Предполагаемый сайт распознавания для MHP14+ представляет собой полинуклеотид размером 22 п.о., расположенный и имеющий следующую последовательность:
(SEQ ID NO: 14) CAAACAGATTCACGTCAGATTT.
ПРИМЕР 2
Продуцирование белка мегануклеазы в E. coli
С целью получения очищенного белка для in vitro анализов активности расщепления мегануклеазой геномной и плазмидной ДНК, фрагменты ДНК, соответствующие открытым рамкам считывания мегануклеазы Lig3-4 (SEQ ID NO: 2) и мегануклеазы MHP14+ (SEQ ID NO: 4), помещали в вектор экспрессии pQE80 (Qiagen), трансформировали в клетки E. coli BL21-Gold (Agilent Technologies) и выращивали в течение ночи на твердой среде LB, содержащей 100 ppm (частей на миллион) карбенициллина. Колонии ресуспендировали в 2 мл среды 2XYT и 250 мкл клеточной суспензии использовали для инокуляции 50 мл культуры 2XYT, дополненной 100 ppm карбенициллина. Культуры выращивали при 37°C в течение 1-1,5 часа или до достижения OD600 0,8, а затем экспрессию белка индуцировали добавлением 0,5 мл 100 мМ IPTG. Культуры охлаждали до комнатной температуры и обеспечивали возможность экспрессии белка в течение 2 часов. Клетки осаждали центрифугированием в течение 10 минут при 5000 rcf (единиц центробежного ускорения). Супернатант сливали, осадок ресуспендировали в 1 мл буфера 1 (50 мМ Tris-HCl (pH 8,0), 500 мМ NaCl, 10 мМ имидазол) и переносили в 1,5 мл микроцентрифужную пробирку. Клетки разрушали путем ультразвуковой обработки в два этапа 1/8'' микронаконечником с 20 импульсами (рабочий цикл 50, мощность 4) на аналоговом устройстве для ультразвуковой обработки Branson 450 Analog Sonifer и центрифугировали при 20000 rcf в течение 15 минут при 4°C. Супернатант разводили 4 мл буфера 1 и загружали на одноразовую колонку, содержащую 0,3 мл никель-содержащей смолы Nickel-NTA Superflow resin (Qiagen). Колонку промывали 5 мл буфера 2 (50 мМ Tris-HCl (pH 8,0), 500 мМ NaCl, 60 мМ имидазол) и белок элюировали 0,6 мл буфера 4 (50 мМ Tris-HCl (pH 8,0), 500 мМ NaCl, 250 мМ имидазол) в колонку Vivaspin (GE). Для концентрирования образцов колонки vivaspin центрифугировали при 14800 rcf в течение примерно 6 минут или до того, как мениск находился между 75 и 50. Замену буфера осуществляли с использованием колонки для обессоливания Zeba Spin Desalting Column (Pierce), предварительно уравновешенной буфером для хранения (25 мМ Tris-HCl (pH 8,0), 100 мМ NaCl, 10 мМ MgCl2, 5 мМ EDTA, 50% глицерин). После замены буфера бычий сывороточный альбумин добавляли до конечной концентрации 100 нг/мкл и очищенный белок хранили при -20°C до применения.
ПРИМЕР 3
In vitro анализы расщепления геномной ДНК
Для создания материала для захвата геномных вариантных сайтов распознавания in vitro анализы проводили с 114 нМ очищенного белка мегануклеазы, выделенного, как описано в примере 2, и 6,07 мкг очищенной геномной ДНК маиса при 32°C в течение 80 минут в конечном объеме 80 мкл в присутствии буфера для расщепления (50 мМ Tris-HCl (pH 7,9), 100 мМ NaCl, 10 мМ MgCl2, 1 мМ DTT, 5 мМ EDTA). После 80 минут всю реакцию останавливали равным объемом стоп-буфера (100 мМ Tris-HCl (pH 8,0), 600 мМ NaCl, 2% SDS, 100 мМ EDTA, 1 мг протеиназы K на мл) и инкубировали при 50°C в течение 30-45 минут. Реакционную смесь с остановленной реакцией очищали с помощью экстракции фенолом/хлороформом и осаждали этанолом в присутствии 0,2 М NaCl. Осажденную геномную ДНК дважды промывали 70% этанолом, высушивали и ресуспендировали в 34 мкл воды.
Концентрацию белка мегануклеазы определяли визуально в гелях Nu-PAGE (Life Technologies) путем расчета интенсивности полосы и последующего ее сравнения с таковой для образцов известной концентрации в серийном разведении, и концентрацию геномной ДНК определяли с использованием флуориметрического анализа с красителем Хехста.
Для подтверждения расщепления (представляющего собой % потери сайтов распознавания мегануклеазой) в предполагаемом геномном сайте распознавания проводили ПЦР в реальном времени на 1 мкл очищенной геномной ДНК с анализом TaqMan, охватывающем сайт распознавания мегануклеазой. % расщепления или потери сайтов распознавания мегануклеазой рассчитывали с помощью способа ΔΔCt относительно внутреннего контроля в анализе TaqMan с использованием пустого контроля в качестве калибровочного стандарта.
ПРИМЕР 4
Улавливание геномных вариантных сайтов распознавания и получение библиотек для глубокого секвенирования Illumina
В данном способе используется подход с новым адаптером, специально подобранным для улавливания расщепленных хоминг-эндонуклеазой I-CreI или сконструированной I-CreI геномных вариантных сайтов распознавания, последовательность которых неизвестна и отличается по составу от предполагаемого сайта распознавания, который отличается от способов с использованием рестрикционных ферментов для осуществления секвенирования с уменьшенным представлением, глубокое секвенирование ДНК, связанное с рестрикцией (RAD-tag или RADseq), полногеномное секвенирование (WGS) или генотипирование с помощью секвенирования (GBS).
Поскольку хоминг-нуклеаза I-CreI образует 4-нуклеотидный 3'-липкий конец в центре своего сайта распознавания длиной 22 п.о. (+2, +1, -1, -2) при расщеплении (Thompson et al. (1992) Gene 119:247-51; и Durrenberger et al. (1993) Mol. Gen. Genet. 236:409-14), и, как было продемонстрировано, расщепляет свой сайт распознавания в контексте различных комбинаций центральных 4 пар оснований (+2, +1, -1, -2) (Molina et al. (2012) Nucleic Acids Res. 40:6936-45), создавали адаптеры, содержащие 4-нуклеотидный 3'-липкий конец, содержащий все возможные комбинации нуклеотидов ДНК (G, T, A или C) липкого конца в эквимолярном распределении. Таким образом, обеспечение эффективного лигирования и полной комплементарности всем возможным липким концам, созданным при расщеплении сайта распознавания в геноме, подвергшемся расщеплению хоминг-эндонуклеазой I-CreI или сконструированной I-CreI.
Подобные стратегии можно использовать для других редкощепящих средств для индукции двунитевого разрыва, таких как нуклеазы "цинковые пальцы" и нуклеазы TALEN, которые расщепляют ДНК неспецифическим каталитическим доменом FokI, создавая липкие концы с вариабельными длиной и нуклеотидным составом в лежащем между ними спейсерном участке (Smith et al. (2000) Nucleic Acids Res. 28:3361-69; и Li et al. (2011) Nucleic Acids Res. 39:359-72). Для улавливания геномных вариантных сайтов распознавания нефосфорилированные биотинилированные адаптеры синтезировали и очищали с помощью HPLC (Integrated DNA Technologies, Inc.), содержащей полностью вырожденный 4-нуклеотидный 3'-нуклеотидный липкий конец, комплементарный 4-нуклеотидному 3'-липкому концу, созданному при расщеплении сайта распознавания мегануклеазой (SEQ ID NO: 5), и лигировали примерно с 2 мкг расщепленной мегануклеазой геномной ДНК (полученной, как описано в примере 3) в 100 мкл реакционной смеси с лигазой T4 (NEB) (представляя первый адаптер в способе для идентификации вариантного сайта распознавания для редкощепящего сконструированного средства для индукции двунитевого разрыва). Образцы, содержащие лигированную ДНК, затем загружали в микропробирки для ультразвуковой обработки и произвольным образом разрезали до среднего размера пика 300 п.о. путем ультразвуковой обработки в системе Covaris E220. Настройки были следующими: 10% рабочий цикл, 140 пиковая мощность падающей волны и 200 циклов на разрыв. Фрагменты с длиной в диапазоне от 200 до 500 п.о. фракционировали с помощью электрофореза в агарозном геле с последующей экстракцией из геля с использованием набора для экстракции из геля Qiagen Gel Extraction Kit в соответствии с рекомендациями производителя.
Небиотинилированные концы подвергали репарации с использованием набора для репарации End-It End repair kit (Epicentre) в 75 мкл реакционной смеси и очищали на колонке (Qiagen). Удлинение 3'-липкого конца на один A осуществляли путем инкубирования репарированной ДНК при 37°C в течение 30 минут в 50 мкл реакционной смеси, содержащей ATP, 1× буфер Кленова (NEBnext) и 15 единиц фрагмента Кленова (без экзонуклеазной активности). Образцы затем очищали с помощью колонки (Qiagen) и лигировали в индексированные TruSeq-совместимые адаптеры Illumina (представляющие собой второй набор адаптеров в способе для идентификации вариантного сайта распознавания для редкощепящего сконструированного средства для индукции двунитевого разрыва) в 50 мкл реакционной смеси, содержащей 0,3 мМ индексированный адаптер, 1× буфер для лигирования Quick ligation buffer и 5 единиц ДНК лигазы T4 (NEB) при комнатной температуре. После лигирования образцы инкубировали при 65°C в течение 15 минут и объем доводили до 100 мкл. Магнитное улавливание с помощью стрептавидина осуществляли с использованием стрептавидиновых гранул Dynabeads M-280 (Invitrogen). Всего 100 мкл ресуспендированных Streptavidin-Dynabeads (M-280) дважды промывали в TE и ресуспендировали в 100 мкл 2X B&W буфера (10 мМ Tris-HCL, 1 мМ EDTA, 100 мкл 0,5 M EDTA, 2 M NaCl). Образцы инкубировали при 30°C в течение 30 минут, супернатант удаляли и гранулы промывали 4 раза 1 мл 1X B&W буфера. Конечный обогащенный образец ресуспендировали в 30 мкл буфера EB.
Фрагменты извлекали из гранул с помощью 12-цикловой ПЦР с использованием мастер-микса Phusion (NEB) в 50 мкл реакционной смеси с в присутствии 0,4 пмоль праймера A для извлечения (5'GTTGACATGCTGGATTGAGACTTC; SEQ ID NO: 6) и праймера B для извлечения (5'CAAGCAGAAGACGGCATACGA; SEQ ID NO: 7) в соответствии с инструкциями производителя, за исключением того, что использовали температуру отжига 66°C и время удлинения 30 секунд. Извлеченную ДНК расщепляли SbfI (NEB) и очищали дважды с гранулами Agencourt AMPure XP Beads (SPRI) в соответствии с инструкциями производителя. Образец лигировали с Illumina-совместимым адаптером с SbfI-совместимым липким концом (SEQ ID NO: 8). Супернатант очищали дважды с использованием гранул Agencourt AMPure XP Beads, вначале с использованием соотношения образца к гранулам 1:1,8, а затем 1:1. Конечные образцы ресуспендировали в 50 мкл буфера EB. Проводили вторую амплификацию со стандартным коктейлем ПЦР праймеров TruSeq PCR (Illumina) с использованием температуры отжига 60°C, с последующей двукратной очисткой с использованием гранул Agencourt AMPure XP Beads при соотношении образца к гранулам 1:1,8, а затем 1:1. Конечный образец ресуспендировали в 20 мкл. Образцы оценивали на биоанализаторе, подвергали относительному количественному анализу с использованием qPCR с праймерами для qPCR Illumina и собирали в пул. Перед секвенированием пулы подвергали селекции по размеру с использованием лабораторного чипа для кристаллографии Xchip (Caliper) в соответствии с инструкциями производителя.
ПРИМЕР 5
Нефосфорилированные адаптеры усиливают обогащение расщепленными мегануклеазой геномными сайтами распознавания
Для оценки эффекта, который фосфорилирование оказывает на способность первого адаптера (который описан в примере 4) к улавливанию и обогащению в отношении расщепленных мегануклеазой геномных сайтов распознавания, были получены библиотеки как с фосфорилированными, так и нефосфорилированными адаптерами. После нормализации концентрации ДНК библиотек их оценивали в отношении обогащения расщепленным предполагаемым сайтом распознавания с помощью ПЦР в реальном времени с анализом TaqMan, непосредственно граничащим с предполагаемым сайтом распознавания. Как показано на фигуре 7, обе библиотеки демонстрировали обогащение по сравнению с пустым контролем, но на графиках амплификации от библиотеки, полученной с нефосфорилированным адаптером, логарифмическая амплификация достигалась на гораздо более раннем цикле амплификации, чем у библиотеки, полученной с фосфорилированным адаптером. При использовании стандартных кривых, полученных из геномной ДНК маиса, библиотеку, полученную с нефосфорилированным адаптером, оценивали как примерно в 900 раз более обогащенную в отношении левой половины предполагаемого сайта распознавания, чем библиотеку, полученную с фосфорилированным адаптером.
ПРИМЕР 6
Глубокое секвенирование Illumina и подрезание после обработки
После улавливания и обогащения в отношении геномных вариантных сайтов распознавания и получения ДНК, как описано в примере 4, создание кластеров и секвенирование рида спаренного конца осуществляли на приборе Illumina cBot и геномном анализаторе Genome Analyzer IIx, соответственно, в соответствии с инструкциями производителя. Примерно 30% (об./об.) контроля phiX ДНК (Illumina) добавляли к раствору библиотеки ДНК перед выделением кластеров. Ожидалось, что произвольный состав оснований фрагментов phiX ДНК компенсирует любое отклонение в составе оснований вблизи сайта для мегануклеазы. Наборы команд на 100 циклов для спаренных концов использовали на геномном анализаторе Illumina Genome Analyzer. Последовательности и оценки качества получали с помощью программного обеспечения Illumina pipeline версии 2.9 для анализа изображения и распознавания оснований. В ходе распознавания оснований данные контроля phiX использовали для получения оценок погрешности и повторного калибрования необработанных оценок качества для других образцов. После первоначального распознавания оснований осуществляли дополнительную фильтрацию с помощью программного обеспечения Illumina, при которой риды исключали, если оценка шума превышает пороговые значения, определенные при распознавании оснований с помощью Illumina pipeline. За превращением результатов распознавания оснований в формат FASTQ (с использованием программного обеспечения Illumina CASSAVA) следовала дополнительная фильтрация, при которой риды подрезали и фильтровали в соответствии с соответствующей оценкой качества для каждого основания (где основания с качественной оценкой ниже 10 подрезали с 3'-конца рида).
ПРИМЕР 7
In silico обогащение и нанесение на физическую карту данных о последовательности относительно геномного стандарта
Для дополнительного обогащения в отношении расщепленных мегануклеазой и лигированных с адаптером фрагментов геномной ДНК набор ридов из эксперимента по секвенированию фильтровали с использованием оригинального сценария, который ищет пары ридов или одиночные объекты, где по меньшей мере один член из пары (или одиночный объект) содержит маркерную последовательность, GCAGGACGT (SEQ ID NO: 9), в начале рида или ее комплементарную последовательность, ACGTCCTGC (SEQ ID NO: 10), в конце рида. Пары или одиночные объекты ридов, совпадающие с этой последовательностью, записывали в новый файл для использования на фазе нанесения на карту, тогда как остальные отбрасывали.
Для воссоединения обеих половин расщепленного геномного сайта распознавания набор обогащенных ридов наносили на физическую карту относительно целевого стандартного генома с использованием программы bowtie версии 0.12.7 (Langmead B, Trapnell C, Pop M, Salzberg SL. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol 10:R25.) для идентификации положений в геноме, гомологичных ридам. Настройки выравнивания для специфичности корректировали индивидуально в каждом конкретном случае в зависимости от характеристик целевого генома и от сходства этого генома с данными исходного генотипа. Полученные в результате выравнивания затем использовали для выявления пика и идентификации вариантных последовательностей распознавания, присутствующих в геноме.
ПРИМЕР 8
Выявление пика и идентификация геномного вариантного сайта распознавания и композиция
С использованием геномных выравниваний, полученных в примере 7, выявление пика выполняли с использованием алгоритма MAC (модельный анализ ChiP-Seq) для выявления пика в Genedata Expressionist Refiner 7.5 (Genedata) с параметрами, перечисленными в таблице 1.
Настройки в алгоритме MAC (модельный анализ ChiP-Seq) для выявления пика в Genedata Expressionist Refiner 7.5 (Genedata)
Хромосомные участки со значительным обогащением по сравнению с пустым контролем экспортировали в excel. Участкам с наибольшим различием между обработанными образцами и пустым контролем отдавали предпочтение, и их подтверждали как обогащенные по сравнению с пустым контролем и имеющие сигнатуру пика, являющуюся результатом расщепления геномной ДНК, в функциональном компоненте Genome Browser в Genedata Expressionist Refiner 7.5. Как показано на фигуре 1, сигнатуры пика для сайта распознавания содержат данные о последовательности, происходящие из и расходящиеся с сайтом расщепления, причем перекрывающийся центр соответствует липкому концу, созданному средством для индукции двунитевого разрыва. Исходя из липкого конца, определенного по сигнатуре пика, можно идентифицировать точную последовательность вариантного сайта распознавания, присутствующую в геномной ДНК, как показано на фигуре 2. Для определения правильной ориентации последовательностей геномного вариантного сайта распознавания, их выравнивали с предполагаемым целевым сайтом как в смысловой, так и в антисмысловой ориентации, при этом наиболее соответствующую ориентацию использовали в качестве последовательности сайта распознавания. Небольшая доля геномных вариантных сайтов распознавания, одинаково хорошо соответствующих с в обеих ориентациях. Их оставляли в смысловой ориентации. Ориентированные геномные вариантные сайты распознавания выравнивали и процентный состав нуклеотидов ДНК рассчитывали для каждого отдельного положения сайта распознавания и сравнивали с предполагаемым сайтом распознавания. Как показано на фигуре 3, некоторые положения в сайтах распознавания проявляли предпочтение к посторонним нуклеотидам; предпочтение в отношении нуклеотида, отличного от того, на который происходит нацеливание в предполагаемом сайте распознавания.
ПРИМЕР 9
In vitro анализы расщепления плазмидной ДНК
Активность расщепления мегануклеазой in vitro также можно анализировать с использованием плазмидной ДНК. Для сравнения активности расщепления мегануклеазой в предполагаемых и вариантных сайтах распознавания гибридизированные олигонуклеотиды (синтезированные Integrated DNA Technologies, Inc.), содержащие предполагаемый или вариантный сайт распознавания с липкими концами EcoRI и HindIII, клонировали в сайты рестрикционных эндонуклеаз HindIII и EcoRI в плазмиде pBluescript SK+ (Stratagene, сейчас компания Agilent Technologies) и анализировали in vitro активность расщепления ДНК, как описано в примере 3 со следующими модификациями. Хронометрированные расщепления осуществляли с 0,25 нМ субстрата-линеаризованной плазмиды, содержащей один предполагаемый или вариантный сайт распознавания, с 25 нМ очищенного белка мегануклеазы. In vitro анализы проводили при 37°C, 28°C и 23°C для наилучшей оценки активности расщепления в заданном вариантном сайте распознавания. Реакционные смеси после остановки реакций очищали с помощью колонки для очистки ПЦР продуктов Qiagen PCR purification column в соответствии с инструкциями производителя и очищенную ДНК разводили в 200 раз перед количественным определением активности расщепления или % потери сайтов распознавания с помощью qPCR.
ПРИМЕР 10
Идентификация вариантных сайтов распознавания и влияние предпочтений к посторонним нуклеотидам на активность расщепления мегануклеазой
Для оценки влияния, которое предпочтения к посторонним нуклеотидам, оказывают на активность расщепления мегануклеазой, предпочтения к посторонним нуклеотидам вводили в предполагаемый сайт распознавания по отдельности и в комбинации (см. фигуру 4), таким образом создавая вариантные сайты распознавания. Примеры таких вариантных сайтов распознавания показаны в таблице 2 для мегануклеазы LIG3-4 и в таблице 3 для мегануклеазы MHP14+.
Перечень вариантных сайтов распознавания мегануклеазы LIG3-4. Нуклеотиды, помещенные в клетки с белой заливкой, указывают на модификацию по сравнению с нуклеотидом в соответствующем расположении в предполагаемом сайте распознавания (SEQ ID NO:13)
Перечень вариантных сайтов распознавания мегануклеазы MHP14+. Нуклеотиды, помещенные в клетки с белой заливкой, указывают на модификацию по сравнению с нуклеотидом в соответствующем расположении в предполагаемом сайте распознавания (SEQ ID NO:14)
Вариантные сайты распознавания затем клонировали в сайты для рестрикционных эндонуклеаз HindIII и EcoRI в векторе pBluescript SK+ и мегануклеазную активность анализировали путем определения % расщепления или % потери сайтов распознавания на субстратах-плазмидной ДНК, как описано в примере 9.
При введении по отдельности в их соответствующие предполагаемые сайты распознавания Lig3-4 и MHP14+ предпочтения к посторонним нуклеотидам придавали активность расщепления плазмидной ДНК равную или большую, чем предполагаемый сайт распознавания (см. фигуру 5A и 5B). Хотя не предпочитаемые нуклеотиды не расщеплялись хорошо (см. фигуру 5A и 5B). Интересно, что даже предпочтения к посторонним нуклеотидам в пределах центральных 4 оснований (+2, +1, -1, -2), как сообщалось, не были непосредственно связаны с повышенной активностью расщепления хоминг-эндонуклеазой I-CreI (Jurica et al. (1998) Mol. Cell 2:469-76; Grizot et al. (2011) Nucleic Acids Res. 39:6124-36, и Ulge et al. (2011) Nucleic Acids Res. 39:4330-9)). Одним исключением из этого была слабая активность расщепления для аденина в положении +2 для мегануклеазы MHP14+. Тем не менее, если все предпочтительные нуклеотиды для MHP14+ оценивали в комбинации, как показано на фигуре 6B (сайты распознавания -3A, -2G, -1T, +2A, +7T, +8G, +11G и -3A, -2G, -1T, +2A, +7T, +8G, +11A), наблюдали активность расщепления, значительно большую таковой в предполагаемом сайте распознавания. Также, если +2A удаляли как в сайте распознавания -3A, -2G, -1T, +7T, +8G, +11G, присутствовало небольшое повышение активности расщепления (см. фигуру 6B), указывая на то, что расщепление по аденину в положении +2 является контекст-специфическим и зависит от распознавания или конформации нуклеотидных оснований, прилегающих к нему.
В комбинации, предпочтения к посторонним нуклеотидам оказывают аддитивное влияние на эффективность расщепления, причем наилучшие комбинации расщепляются примерно в 5-6 раз более эффективно, чем предполагаемый сайт распознавания (фигура 6). Многие из вариантных сайтов распознавания можно даже расщеплять при температурах до 23°C в случае, когда только небольшая активность расщепления наблюдалась в предполагаемом сайте распознавания (фигура 6A и B). Некоторые комбинации предпочтительных посторонних нуклеотидов в 4 центральных основаниях (+2,+1,-1,-2) и в непосредственно прилегающих положениях (+3,-3) влияли на величину приобретенной активности расщепления в сравнении с предполагаемым сайтом распознавания.
Взятые в совокупности, данные авторов настоящего изобретения указывают на то, что способы, описанные в данном документе, можно применять для выведения предпочтительных контактов оснований ДНК, образуемых мегануклеазой в отдельных положениях вдоль ее ДНК-связывающей поверхности контакта, обеспечивая тщательную оценку специфичности расщепления, обеспечивая новый подход к изучению специфичности мегануклеазы в контексте геномной ДНК. Способы авторов настоящего изобретения также позволяют идентификацию вариантных сайтов распознавания, которые расщепляются более эффективно, чем предполагаемый сайт распознавания.
ПРИМЕР 11
Применения вариантных сайтов распознавания у растения или животных
Вариантные сайты распознавания, идентифицированные в примере 10, с улучшенной активностью расщепления по сравнению с предполагаемым сайтом распознавания (-7C, 8T (Lig3-4); -11C, -7C, -2G, -1T, +8T (Lig3-4); -11C, -7C, -1T, +8T (Lig3-4); -3A, -2G, -1T, +2A, +7T, +8G, +11G (MHP14+); -3A, -2G, -1T, +2A, +7T, +8G, +11A (MHP14+); -3A, -2G, -1T, +7T, +8G, +11G (MHP14+); -2G, -1T, +2A, +7T, +8G, +11G (MHP14+); -2G, -1T, +7T, +8G, +11G (MHP14+), соответствующие SEQ ID NO: 13-35), или любой другой вариантный сайт распознавания, идентифицированные с помощью способа, описанного в данном документе, можно трансформировать в геном любого растения или животного и подвергнуть целенаправленному воздействию для мутагенеза или вставки гена. Поскольку активность расщепления в этих сайтах распознавания является повышенной в сравнении с предполагаемым сайтом распознавания, также можно повысить и частоты модификации сайта, в том числе делеции, вставки или любой комбинации этих двух модификаций. Вариантные сайты распознавания также можно расположить по отдельности или в комбинации на трансгенных кассетах экспрессии, обеспечивая возможность изменения, вырезания или вставки трансгенных частей.
ПРИМЕР 12
Применение к другим редкощепящим реактивам для индукции двунитевого разрыва
Поскольку олигонуклеотиды как с 5'-, так и с 3'-вырожденными концами можно синтезировать в широком диапазоне специфических для пользователя конфигураций и значений длины (Integrated ДНК Technologies, Inc) и гибридизовать с образованием двунитевых ДНК адаптеров либо с 5'-, либо с 3'-вырожденным липким концом, установленные в данном документе способы могут быть применимы к любому редкощепящему реактиву для индукции двунитевого разрыва, который создает липкий конец из оснований ДНК при расщеплении. Они будут включать, но не ограничиваются другими хоминг-эндонуклеазами, нуклеазами "цинковые пальцы" и TALEN.
ПРИМЕР 13
Трансформация незрелых зародышей маиса
Трансформация может быть осуществлена с помощью различных способов, о которых известно, что они являются эффективными в растениях, включая опосредованную частицами доставку, опосредованную Agrobacterium трансформацию, опосредованную PEG доставку и электропорацию.
a. Опосредованная частицами доставка
Трансформацию незрелых зародышей маиса при помощи опосредованной частицами доставки проводили следующим образом. Составы сред представлены ниже.
Початки очищали от листовой обвертки и подвергали поверхностной стерилизации в 30% отбеливателе Clorox вместе с 0,5% моющего средства Micro в течение 20 минут, и ополаскивали дважды стерильной водой. Незрелые зародыши выделяли и помещали рубчиком вниз (щитком вверх), 25 зародышей на планшет, в среду 560Y на 4 часа и затем выравнивали в пределах 2,5-см зоны-мишени в препарате для бомбардировки. Альтернативно, выделенных зародышей помещали в 560L (среда для инициирования) и помещали в темноту при температурах, варьирующих в диапазоне от 26°C до 37°C, на 8-24 часа перед помещением в 560Y на 4 часа при 26°C перед бомбардировкой, как описано выше.
Плазмиды, содержащие ген средства, индуцирующего двунитевой разрыв, и донорную ДНК, конструировали при помощи стандартных методик молекулярной биологии и подвергали совместной бомбардировке с плазмидами, содержащими контролирующие развитие гены ODP2 (фактор транскрипции ODP2 с доменами AP2 (отвечающий за развитие семязачатка белок 2); US20090328252 A1) и Wushel (US2011/0167516).
Плазмиды и ДНК, представляющую интерес, осаждали на гранулах золота размером 0,6 мкм (средний диаметр) при помощи водорастворимого катионного липида Tfx™-50 (№ по кат. E1811, Promega, Мэдисон, Висконсин, США) следующим образом. Раствор ДНК готовили на льду с применением 1 мкг плазмидной ДНК и необязательно других конструкций для совместной бомбардировки, таких как 50 нг (0,5 мкл) каждой плазмиды, содержащей контролирующие развитие гены ODP2 (фактор транскрипции ODP2 с доменами AP2 (отвечающий за развитие семязачатка белок 2); US20090328252 A1) и Wushel. К предварительно смешанной ДНК добавляли 20 мкл подготовленных частиц золота (15 мг/мл) и 5 мкл TFX-50 в воде и осторожно перемешивали. Частицы золота осаждали в микроцентрифуге при 10000 об./мин. в течение 1 мин. и супернатант удаляли. Полученный в результате осадок осторожно ополаскивали с помощью 100 мл 100% EtOH без ресуспендирования осадка и промывочный EtOH осторожно удаляли. Добавляли 105 мкл 100% EtOH и частицы ресуспендировали путем непродолжительной ультразвуковой обработки. Затем 10 мкл наносили на центральную часть каждого макроносителя и им позволяли высохнуть приблизительно за 2 минуты до бомбардировки.
Альтернативно, плазмиды и ДНК, представляющую интерес, осаждали на гранулах вольфрама размером 1,1 мкм (средний диаметр) при помощи процедуры осаждения хлоридом кальция (CaCl2) путем смешивания 100 мкл подготовленных частиц вольфрама в воде, 10 мкл (1 мкг) ДНК в буфере Tris-EDTA (1 мкг общей ДНК), 100 мкл 2,5 M CaCl2 и 10 мкл 0,1 M спермидина. Каждый реактив добавляли последовательно к суспензии частиц вольфрама с перемешиванием. Конечную смесь подвергали непродолжительной ультразвуковой обработке и обеспечивали инкубацию при постоянном перемешивании на вихревой мешалке в течение 10 минут. После периода осаждения пробирки центрифугировали в течение непродолжительного времени, жидкость удаляли, а частицы промывали 500 мл 100% этанола с последующим центрифугированием в течение 30 секунд. Жидкость вновь удаляли и к конечному осадку частиц вольфрама добавляли 105 мкл 100% этанола. Для бомбардировки генной пушкой частицы вольфрама с ДНК подвергали непродолжительной ультразвуковой обработке. 10 мкл частиц вольфрама с ДНК наносили на центральную часть каждого макроносителя, после чего нанесенным частицам позволяли высохнуть приблизительно за 2 минуты до бомбардировки.
Планшеты с образцами бомбардировали на уровне №4 с помощью гелиевой генной пушки Biorad. Все образцы получали однократный выстрел при 450 фунт/кв.дюйм, при этом общее количество аликвот, взятых из каждой пробирки с подготовленными частицами с ДНК, составляло десять.
После бомбардировки зародыши инкубировали в 560P (поддерживающей среде) в течение 12-48 часов при температурах, варьирующих в диапазоне от 26°C до 37°C, а затем помещали в условия температуры 26°C. Через 5-7 дней зародыши переносили в селективную среду 560R, содержащую 3 мг/литр биалафоса, и субкультивировали каждые 2 недели при 26°C. Через приблизительно 10 недель отбора каллюсные клоны, устойчивость которых была выявлена в результате отбора, переносили на среду 288J для инициирования регенерации растений. После созревания соматических зародышей (2-4 недели) хорошо развитые соматические зародыши переносили в среду для прорастания и переносили в освещаемую комнату для культивирования. Спустя приблизительно 7-10 дней развивающиеся саженцы переносили в безгормональную среду 272V в пробирки на 7-10 дней, пока саженцы хорошо укоренятся. Растения затем переносили во вставки в лотках (эквивалентные горшку на 2,5 дюйма), содержащих почвенную горшечную смесь, и выращивали в течение 1 недели в вегетационной камере, впоследствии выращивая в течение дополнительных 1-2 недель в теплице, перенося затем в горшки Classic 600 (1,6 галлона) и выращивая до зрелости. За растениями следили и оценивали в баллах по эффективности трансформации и/или модификации регенеративных способностей.
Среда для инициирования (560L) содержит 4,0 г/л основных солей N6 (SIGMA C-1416), 1,0 мл/л витаминной смеси Эрикссона (1000X SIGMA-1511), 0,5 мг/л тиамина-HCl, 20,0 г/л сахарозы, 1,0 мг/л 2,4-D и 2,88 г/л L-пролина (доведенных до объема с помощью D-I H2O после доведения pH до 5,8 с помощью KOH); 2,0 г/л Gelrite (добавленного после доведения до объема с помощью D-I H2O) и 8,5 мг/л нитрата серебра (добавленного после стерилизации среды и охлаждения до комнатной температуры).
Поддерживающая среда (560P) содержит 4,0 г/л основных солей N6 (SIGMA C-1416), 1,0 мл/л витаминной смеси Эрикссона (1000X SIGMA-1511), 0,5 мг/л тиамина-HCl, 30,0 г/л сахарозы, 2,0 мг/л 2,4-D и 0,69 г/л L-пролина (доведенных до объема с помощью D-I H2O после доведения pH до 5,8 с помощью KOH); 3,0 г/л Gelrite (добавленного после доведения до объема с помощью D-I H2O) и 0,85 мг/л нитрата серебра (добавленного после стерилизации среды и охлаждения до комнатной температуры).
Среда для бомбардировки (560Y) содержит 4,0 г/л основных солей N6 (SIGMA C-1416), 1,0 мл/л витаминной смеси Эрикссона (1000X SIGMA-1511), 0,5 мг/л тиамина-HCl, 120,0 г/л сахарозы, 1,0 мг/л 2,4-D и 2,88 г/л L-пролина (доведенных до объема с помощью D-I H2O после доведения pH до 5,8 с помощью KOH); 2,0 г/л Gelrite (добавленного после доведения до объема с помощью D-I H2O) и 8,5 мг/л нитрата серебра (добавленного после стерилизации среды и охлаждения до комнатной температуры).
Селективная среда (560R) содержит 4,0 г/л основных солей N6 (SIGMA C-1416), 1,0 мл/л витаминной смеси Эрикссона (1000X SIGMA-1511), 0,5 мг/л тиамина-HCl, 30,0 г/л сахарозы и 2,0 мг/л 2,4-D (доведенных до объема с помощью D-I H2O после доведения pH до 5,8 с помощью KOH); 3,0 г/л Gelrite (добавленного после доведения до объема с помощью D-I H2O) и 0,85 мг/л нитрата серебра и 3,0 мг/л биалафоса (оба из которых добавлены после стерилизации среды и охлаждения до комнатной температуры).
Среда для регенерации растений (288J) содержит 4,3 г/л солей по MS (GIBCO 11117-074), 5,0 мл/л исходного раствора витаминов по MS (0,100 г никотиновой кислоты, 0,02 г/л тиамина-HCL, 0,10 г/л пиридоксина-HCL и 0,40 г/л глицина, доведенных до объема с помощью очищенной D-I H2O) (Murashige and Skoog (1962) Physiol. Plant. 15:473), 100 мг/л миоинозита, 0,5 мг/л зеатина, 60 г/л сахарозы и 1,0 мл/л 0,1 мМ абсцизовой кислоты (доведенных до объема очищенной D-I H2O после доведения pH до 5,6); 3,0 г/л Gelrite (добавленного после доведения до объема с помощью D-I H2O), а также 1,0 мг/л индолилуксусной кислоты и 3,0 мг/л биалафоса (добавленных после стерилизации среды и охлаждения до 60ºC).
Безгормональная среда (272V) содержит 4,3 г/л солей по MS (GIBCO 11117-074), 5,0 мл/л исходного раствора витаминов по MS (0,100 г/л никотиновой кислоты, 0,02 г/л тиамина-HCl, 0,10 г/л пиридоксина-HCl и 0,40 г/л глицина, доведенных до объема с помощью очищенной D-I H2O), 0,1 г/л миоинозит и 40,0 г/л сахарозы (доведенных до объема с помощью очищенной D-I H2O после доведения pH до 5,6); и 6 г/л бактоагара (добавленного после доведения до объема с помощью очищенной D-I H2O), ее стерилизовали и охлаждали до 60°C.
b. Опосредованная Agrobacterium трансформация
Опосредованную Agrobacterium трансформацию проводили по сути согласно описанному в Djukanovic et al. (2006) Plant Biotech J 4:345-57. Вкратце, незрелых зародышей в возрасте 10-12 дней (размером 0,8-2,5 мм) вырезали из стерилизованных зерен и помещали в жидкую среду (4,0 г/л основных солей N6 (Sigma C-1416), 1,0 мл/л витаминной смеси Эрикссона (Sigma E-1511), 1,0 мг/л тиамина-HCl, 1,5 мг/л 2,4-D, 0,690 г/л L-пролина, 68,5 г/л сахарозы, 36,0 г/л глюкозы, pH 5,2). После сбора зародышей среду заменяли на 1 мл Agrobacterium в концентрации 0,35-0,45 OD550. Зародыши маиса инкубировали с Agrobacterium в течение 5 мин. при комнатной температуре, затем смесь выливали на планшет со средой, содержащий 4,0 г/л основных солей N6 (Sigma C-1416), 1,0 мл/л витаминной смеси Эрикссона (Sigma E-1511), 1,0 мг/л тиамина-HCl, 1,5 мг/л 2,4-D, 0,690 г/л L-пролина, 30,0 г/л сахарозы, 0,85 мг/л нитрата серебра, 0,1 нМ ацетосирингона и 3,0 г/л Gelrite, pH 5,8. Зародышей инкубировали рубчиком вниз в темноте в течение 3 дней при 20°C, затем инкубировали в течение 4 дней в темноте при 28°C, затем переносили на новые планшеты со средой, содержащей 4,0 г/л основных солей N6 (Sigma C-1416), 1,0 мл/л витаминной смеси Эрикссона (Sigma E-1511), 1,0 мг/л тиамина-HCl, 1,5 мг/л 2,4-D, 0,69 г/л L-пролина, 30,0 г/л сахарозы, 0,5 г/л буфера MES, 0,85 мг/л нитрата серебра, 3,0 мг/л биалафоса, 100 мг/л карбенициллина и 6,0 г/л агара, pH 5,8. Зародыши подвергали субкультивированию каждые три недели до идентификации трансгенных объектов. Соматический эмбриогенез индуцировали путем переноса небольшого количества ткани в среду для регенерации (4,3 г/л солей по MS (Gibco 11117), 5,0 мл/л исходного раствора витаминов по MS, 100 мг/л миоинозита, 0,1 мкМ ABA, 1 мг/л IAA, 0,5 мг/л зеатина, 60,0 г/л сахарозы, 1,5 мг/л биалафоса, 100 мг/л карбенициллина, 3,0 г/л Gelrite, pH 5,6) и инкубации в темноте в течение двух недель при 28°C. Весь материал с видимыми побегами и корнями переносили в среду, содержащую 4,3 г/л солей по MS (Gibco 11117), 5,0 мл/л исходного раствора витаминов по MS, 100 мг/л миоинозита, 40,0 г/л сахарозы, 1,5 г/л Gelrite, pH 5,6, и инкубировали под искусственным освещением при 28°C. Спустя неделю сеянцы перемещали в стеклянные пробирки, содержащие ту же самую среду, и выращивали до отбора образцов из них и/или пересадки их в почву.
ПРИМЕР 14
Транзиентная экспрессия BBM усиливает трансформацию
Параметры протокола трансформации могут быть модифицированы для подтверждения того, что активность BBM является транзиентной. Один такой способ включает осаждение плазмиды, содержащей ВВМ, способом, который позволяет происходить транскрипции и экспрессии, но предотвращает последующее высвобождение ДНК, например, посредством применения химического соединения PEI.
В одном примере плазмиду с BBM осаждали на частицах золота с помощью PEI, тогда как трансгенную кассету экспрессии (UBI::moPAT~GFPm::PinII; moPAT представляет собой оптимизированный для экспрессии в маисе ген PAT), подлежащую интеграции, осаждали на частицах золота при помощи стандартного способа с применением хлорида кальция.
Вкратце, частицы золота покрывали с помощью PEI следующим образом. Вначале частицы золота промывали. Тридцать пять мг частиц золота со средним диаметром 1,0 (A.S.I. № 162-0010) отвешивали в микроцентрифужную пробирку, и добавляли 1,2 мл абсолютного EtOH, и перемешивали вихревой мешалкой в течение одной минуты. Пробирку инкубировали в течение 15 минут при комнатной температуре и затем центрифугировали с высокой скоростью, используя микроцентрифугу, в течение 15 минут при 4°C. Надосадочную жидкость сливали, и добавляли свежую аликвоту этанола (EtOH) на 1,2 мл, перемешивали вихревой мешалкой в течение одной минуты, центрифугировали в течение одной минуты, и надосадочную жидкость снова сливали (это повторяли дважды). Добавляли свежую аликвоту EtOH на 1,2 мл, и эту суспензию (частицы золота в EtOH) хранили при -20°C в течение нескольких недель. Для покрытия частиц полиэтиленимином (PEI; Sigma №P3143) 250 мкл смеси промытых частиц золота/EtOH центрифугировали, и EtOH сливали. Частицы промывали один раз в 100 мкл ddH2O для удаления остаточного этанола, добавляли 250 мкл 0,25 мМ PEI с последующей импульсной ультразвуковой обработкой для суспендирования частиц, и затем пробирку погружали в ванну с сухим льдом/EtOH для мгновенной заморозки суспензии, которую затем лиофилизировали в течение ночи. На данной стадии сухие покрытые частицы могут храниться при -80°C в течение по меньшей мере 3 недель. Перед использованием частицы ополаскивали 3 раза с помощью аликвот 2,5 мМ буфера HEPES на 250 мкл, pH 7,1, с 1× импульсной ультразвуковой обработкой и последующим быстрым перемешиванием вихревой мешалкой перед каждым центрифугированием. Затем частицы суспендировали в конечном объеме 250 мкл буфера HEPES. Аликвоту частиц на 25 мкл добавляли в неиспользованные пробирки перед прикреплением ДНК. Для прикрепления непокрытой ДНК частицы подвергали импульсной ультразвуковой обработке, затем добавляли 1 мкг ДНК (в 5 мкл воды) с последующим перемешиванием путем втягивания и выпуска пипеткой несколько раз с помощью Pipetteman и инкубировали в течение 10 минут. Частицы центрифугировали в течение непродолжительного времени (т.e. 10 секунд), надосадочную жидкость удаляли, и добавляли 60 мкл EtOH. Частицы осажденной с помощью PEI ДНК-1 промывали дважды в 60 мкл EtOH. Частицы центрифугировали, надосадочную жидкость сливали и частицы ресуспендировали в 45 мкл воды. Для прикрепления второй ДНК (ДНК-2) применяли осаждение при помощи TFX-50. 45 мкл суспензии частицы/ДНК-1 подвергали непродолжительной ультразвуковой обработке, и затем добавляли 5 мкл 100 нг/мкл ДНК-2 и 2,5 мкл TFX-50. Раствор помещали на ротационный встряхиватель на 10 минут, центрифугировали при 10000 g в течение 1 минуты. Супернатант удаляли и частицы ресуспендировали в 60 мкл EtOH. Раствор наносили на макроносители, и частицы золота, к которым были последовательно прикреплены ДНК-1 и ДНК-2, доставляли в клетки щитка незрелых зародышей Hi-II на стадии 10 DAP, используя стандартный протокол для PDS-1000. В этом эксперименте плазмида с ДНК-1 содержала кассету экспрессии UBI::RFP::pinII, а плазмида с ДНК-2 содержала кассету экспрессии UBI::CFP::pinII. Через два дня после бомбардировки наблюдали транзиентную экспрессию флуоресцентных маркеров как CFP, так и RFP в виде многочисленных красных и синих клеток на поверхности незрелого зародыша. Зародышей затем помещали в неселективную культуральную среду, и им позволяли расти в течение 3 недель перед оцениванием в баллах стабильных колоний. После этого 3-недельного периода наблюдали 10 многоклеточных, стабильно экспрессирующих синих колоний по сравнению с лишь одной красной колонией. Это показало, что осаждение с помощью PEI можно применять для эффективного введения ДНК для транзиентной экспрессии, при этом существенно снижая степень интеграции вводимой осажденной с помощью PEI ДНК и, таким образом, ослабляя регенерацию трансгенных объектов, экспрессирующих RFP. Таким образом, осаждение с помощью PEI можно применять для обеспечения транзиентной экспрессии BBM и/или WUS2.
Например, частицы вначале покрывали UBI::BBM::pinII при помощи PEI, затем покрывали UBI::moPAT~YFP при помощи TFX-50, а затем вводили в клетки щитка на поверхности незрелых зародышей посредством бомбардировки. Осаждение, опосредованное PEI, обуславливает высокую частоту встречаемости клеток с транзиентной экспрессией на поверхности незрелого зародыша и чрезвычайно низкие частоты регенерации стабильных трансформантов (по сравнению со способом с использованием TFX-50). Таким образом, предполагается, что осажденная с помощью PEI кассета с BBM экспрессируется транзиентно и стимулирует всплеск эмбриогенного роста на бомбардируемой поверхности ткани (т.e. поверхности щитка), но эта плазмида не будет интегрироваться. Предполагается, что плазмида с PAT~GFP, освобожденная от частиц золота, на которых она была осаждена с помощью Ca++, интегрируется и экспрессирует селектируемый маркер с частотой, которая обуславливает существенно улучшенную регенерацию трансгенных объектов. В качестве контрольной обработки осажденные с помощью PEI частицы, содержащие UBI::GUS::pinII (вместо BBM), смешивали с частицами с PAT~GFP, осажденными с помощью Ca++. Незрелых зародышей после обоих обработок перемещали в культуральную среду, содержащую 3 мг/л биалафоса. Предполагают, что GFP+ каллюсы, устойчивые к биалафосу, будут наблюдаться через 6-8 недель с намного более высокой частотой в случае обработки PEI/BBM по сравнению с контрольной обработкой (PEI/GUS).
В качестве альтернативного способа плазмиду с BBM осаждали на частицах золота с помощью PEI и затем вводили в клетки щитка на поверхности незрелых зародышей, и последующая транзиентная экспрессия гена BBM вызывала быструю пролиферацию в ходе роста эмбриогенного каллюса. В течение данного периода индуцированного роста экспланты обрабатывали Agrobacterium, используя стандартные способы для маиса (см. пример 1), с доставкой T-ДНК в клетку посредством ввода трансгенной кассеты экспрессии, такой как UBI::moPAT~GFPm::pinII. После совместного культивирования эксплантам позволяли регенерировать в нормальной культуральной среде, и их затем перемещали в культуральную среду, содержащую 3 мг/л биалафоса. Предполагают, что GFP+ каллюсы, устойчивые к биалафосу, будут наблюдаться через 6-8 недель с намного более высокой частотой в случае обработки PEI/BBM по сравнению с контрольной обработкой (PEI/GUS).
Может быть необходимым "активизировать" рост каллюса путем транзиентной экспрессии BBM и/или полинуклеотидных продуктов WUS2. Это может быть выполнено путем доставки 5'-кэпированной полиаденилированной РНК BBM и WUS2, кассет экспрессии, содержащих ДНК BBM и WUS2, или белков BBM и/или WUS2. Все эти молекулы могут быть доставлены при помощи биолистической генной пушки. Например, 5'-кэппированная полиаденилированная BBM и/или WUS2 РНК может быть легко создана in vitro, применяя набор Ambion's mMessage mMachine. РНК подвергали совместной доставке вместе с ДНК, содержащей полинуклеотид, представляющий интерес, и маркер, используемый для отбора/скрининга, как например, Ubi::moPAT~GFPm::PinII. Предполагается, что клетки, получающие РНК, немедленно начнут делиться быстрее, и большая часть этих клеток будет иметь интегрированный агрономически важный ген. Может быть дополнительно подтверждено, что эти трансгенные объекты представляют собой трансгенные клональные колонии, потому что они будут также экспрессировать белок слияния PAT~GFP (и, таким образом, будут проявлять зеленую флуоресценцию при соответствующем освещении). Растения, регенерированные из данных зародышей, могут затем подвергаться скринингу в отношении наличия полинуклеотида, представляющего интерес.
ПРИМЕР 15
Получение и трансформация в модельной системе
культур соматических зародышей сои векторами экспрессии в сое и регенерация растений
Условия культивирования:
Эмбриогенные суспензионные культуры сои (сорт Jack) поддерживали в 35 мл жидкой среды SB196 (ниже) на ротационном встряхивателе при 150 об./мин. и 26°C c люминесцентными лампами холодного белого света с фотопериодом 16:8 часов день/ночь при интенсивности света 60-85 мкЭрг/м2/с. Культуры пересевали от одного раза в 7 дней до одного раза в две недели путем инокуляции приблизительно 35 мг ткани в 35 мл свежей жидкой SB196 (предпочтительный интервал между пересевами составлял 7 дней).
Эмбриогенные суспензионные культуры сои трансформировали с помощью плазмид экспрессии для сои посредством способа бомбардировки частицами (Klein et al., Nature, 327:70 (1987)) с применением устройства DuPont Biolistic PDS1000/HE (гелиевая модифицированная установка) для всех трансформаций.
Инициация эмбриогенных суспензионных культур сои
Осуществляли инициацию культур сои дважды в месяц с интервалом 5-7 дней между каждой инициацией. Стручки с незрелыми семенами от доступных растений сои собирали через 45-55 дней после посадки. Семена удаляли из стручков и помещали в стерилизованный контейнер Magenta. Семена сои стерилизовали путем встряхивания в течение 15 мин в 5% растворе Clorox с 1 каплей мыла “Ivory” (т.е. 95 мл автоклавированной дистиллированной воды плюс 5 мл Clorox и 1 каплей мыла, тщательно смешано). Семена промывали с использованием 2 1-литровых флаконов со стерильной дистиллированной водой и семена размером менее 4 мм помещали на отдельные предметные стекла микроскопа. Срезали острый конец семени и выдавливали семядоли из семенной оболочки. Когда культуры были подготовлены для проведения трансформации, семядоли переносили на плашки, содержащие среду SB1 (25-30 семядоль на плашку). Планшеты обертывали волокнистой лентой и поддерживали при 26°C с освещением флуоресцентными лампами холодного белого с фотопериодом день/ночь 16:8 часов при интенсивности света 60-80 мкЭрг/м2/с в течение восьми недель с заменой среды каждые 4 недели. Когда культуры были подготовлены для экспериментов в модельной системе, семядоли переносили на плашки, содержащие среду SB199 (25-30 семядоль на плашку), на 2 недели, а затем переносили на SB1 на 2-4 недели. Условия освещения и температуры были такими же, как описано выше. После инкубирования на среде SB1 вторичные зародыши вырезали и помещали в жидкую среду SB196 на 7 дней.
Получение ДНК для бомбардировки
Для бомбардировки использовали либо интактную плазмиду, либо фрагмент ДНК плазмиды, содержащий гены, представляющие интерес, и селектируемый маркерный ген. Фрагменты из плазмид экспрессии в сое получали путем выделения расщепленных плазмид в геле. В каждом случае 100 мкг плазмидной ДНК использовали в 0,5 мл специфической смеси ферментов, описанной ниже. Плазмиды расщепляли AscI (100 единиц) в буфере NEBuffer 4 (20 мМ Tris-ацетат, 10 мМ ацетат магния, 50 мМ ацетат калия, 1 мМ дитиотреитол, pH 7,9), 100 мкг/мл BSA и 5 мМ бета-меркаптоэтаноле при 37°C в течение 1,5 часа. Полученные фрагменты ДНК разделяли с помощью электрофореза в геле на 1% агарозе SeaPlaque GTG (BioWhitaker Molecular Applications) и фрагменты ДНК, содержащие кассеты с генами, вырезали из агарозного геля. ДНК очищали от агарозы с использованием расщепляющего фермента GELase согласно протоколу производителя.
Содержащую 3 мг золотых частиц (3 мг золота) 50 мкл аликвоту стерильной дистиллированной воды добавляли к 30 мкл раствора ДНК в концентрации 10 нг/мкл (либо интактной плазмиды, либо фрагмента ДНК, полученного, как описано в данном документе), 25 мкл 5 M CaCl2 и 20 мкл 0,1 M спермидина. Смесь встряхивали 3 минуты на встряхивателе Vortex при уровне 3 и центрифугировали в течение 10 секунд на настольной микроцентрифуге. Супернатант удаляли с последующим промыванием 400 мкл 100% этанола и еще одного краткого центрифугирования. 400 мкл этанола удаляли и осадок ресуспендировали в 40 мкл 100% этанола. Пять мкл суспензии ДНК распределяли на каждый диск-носитель в устройстве для биолистической бомбардировки Biolistic PDS1000/HE. Каждая 5 мкл аликвота содержала примерно 0,375 мг золота на бомбардировку (например, на диск).
Для трансформаций в модельной системе протокол являлся идентичным за исключением нескольких незначительных изменений (т.е. 1 мг золотых частиц добавляли к 5 мкл 1 мкг/мкл раствору ДНК, использовали 50 мкл 2,5 M CaCl2 и осадок в конечном счете ресуспендировали в 85 мкл 100% этанола, таким образом обеспечивая 0,058 мг золотых частиц на бомбардировку).
Получение тканей и бомбардировка ДНК
Примерно 150-200 мг семидневных эмбриогенных суспензионных культур помещали в пустую стерильную 60×15 мм чашку Петри и чашку накрывали пластмассовой сеткой. Обеспечивали разрежение в камере до пониженного давления 27-28 дюймов ртутного столба и ткань бомбардировали одним или двумя выстрелами на плашку, причем давление разрывам мембраны устанавливали на 1100 фунт/дюйм2. Ткань помещали примерно в 3,5 дюймах от задерживающего/ограничительного экрана. Условия трансформации в модельной системе были идентичными за исключением использования 100-150 мг эмбриогенной ткани, давление разрыва устанавливали на 650 фунт/дюйм2 и ткань помещали примерно в 2,5 дюймах от задерживающего экрана.
Отбор трансформированных зародышей
Отбор трансформированных зародышей производили либо с использованием гигромицина (когда ген гигромицин-B-фосфотрансферазы (HPT) использовали в качестве селектируемого маркера) или хлорсульфурона (когда ген ацетолактат-синтазы (ALS) использовали в качестве селектируемого маркера).
После бомбардировки ткань помещали в свежую среду SB196 и культивировали, как описано выше. Через шесть-восемь дней после бомбардировки SB196 заменяли свежей SB196, содержащей либо 30 мг/л гигромицина, либо 100 нг/мл хлорсульфурона в зависимости от используемого селектируемого маркера. Селективную среду обновляли каждую неделю. Через четыре-шесть недель после отбора наблюдали рост зеленой трансформированной ткани из нетрансформированных некротических эмбриогенных кластеров.
Созревание зародышей
Для получения трансформантов выделенную зеленую ткань удаляли и инокулировали в многолуночные планшеты для образования при помощи клонального размножения новых трансформированных эмбриогенных суспензионных культур. Трансформированные эмбриогенные кластеры культивировали в течение четырех-шести недель в многолуночных планшетах при 26°C в SB196 под флуоресцентными лампами холодного белого света (Phillips cool white Econowatt F40/CW/RS/EW) и лампами Agro (Phillips F40 Agro) (40 Вт) с фотопериодом 16:8 ч. при интенсивности освещения 90-120 мкЭрг/м2с. После этого периода времени зародышевые кластеры удаляли в твердой агаровой среде, SB166, в течение одной-двух недель, а затем субкультивировали в среду SB103 на 3-4 недели для созревания зародышей. После созревания на планшетах в SB103 отдельные зародыши удаляли из кластеров, сушили и подвергали скринингу в отношении изменений в составе их жирных кислот, как описано в примере 7.
Для трансформаций в модельной системе созревание зародышей обеспечивали в жидкой среде для тканевой дифференцировки и созревания сои (жидкая среда SHaM; Schmidt et al., Cell Biology and Morphogenesis 24:393 (2005)) с использованием модифицированной процедуры. Вкратце, после 4 недель отбора в SB196, как описано выше, зародышевые кластеры удаляли в 35 мл SB228 (жидкой среды SHaM) в 250 мл колбу Эрленмейера. Ткань поддерживали в жидкой среде SHaM на ротационном встряхивателе при 130 об./мин. и при 26°C с освещением флуоресцентными лампами холодного белого с фотопериодом день/ночь 16:8 часов при интенсивности света 60-85 мкЭрг/м2/с в течение 2 недель до созревания зародышей. Зародыши выращивали в течение 2 недель в жидкой среде SHaM являлись эквивалентными по размеру и содержанию жирных кислот зародышам, культивируемым на среде SB166/SB103 в течение 5-8 недель.

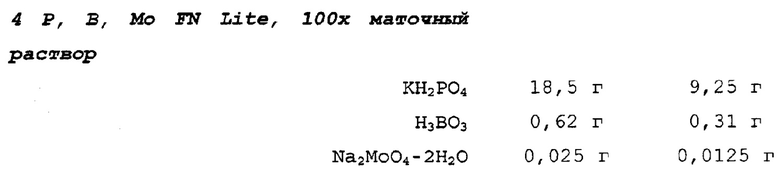
Плотная среда SB1 (на литр)
1 упаковка солей MS (Gibco/ BRL - № в каталоге 11117-066)
1 мл витаминов B5, 1000X маточный раствор
31,5 г глюкозы
2 мл 2,4-D (конечная концентрация 20 мг/л)
pH 5,7
8 г агара TC
Плотная среда SB199 (на литр)
1 упаковка солей MS (Gibco/ BRL - № в каталоге 11117-066)
1 мл витаминов B5, 1000X маточный раствор
30 г сахарозы
4 мл 2,4-D (конечная концентрация 40 мг/л)
pH 7,0
2 г Gelrite
Плотная среда SB 166 (на литр)
1 упаковка солей MS (Gibco/ BRL - № в каталоге 11117-066)
1 мл витаминов B5, 1000X маточный раствор
60 г мальтозы
750 мг гексагидрата MgCl2
5 г активированного угля
pH 5,7
2 г gelrite
Плотная среда SB 103 (на литр)
1 упаковка солей MS (Gibco/ BRL - № в каталоге 11117-066)
1 мл витаминов B5, 1000X маточный раствор
60 г мальтозы
750 мг гексагидрата MgCl2
pH 5,7
2 г gelrite
Плотная среда SB 71-4 (на литр)
1 флакон солей Gamborg’s B5 с сахарозой (Gibco/BRL - № в каталоге 21153-036)
pH 5,7
5 г агара TC
Маточный раствор 2,4-D
Получают предварительно приготовленным от Phytotech № в каталоге D 295 - концентрация 1 мг/мл.
Маточный раствор витаминов B5 (на 100 мл)
Аликвоты хранят при -20°C
10 г миоинозита
100 мг никотиновой кислоты
100 мг пиридоксина-HCl
1 г тиамина
Если раствор не растворяется достаточно быстро, применяют низкий уровень нагрева с помощью горячей плиты для нагрева.
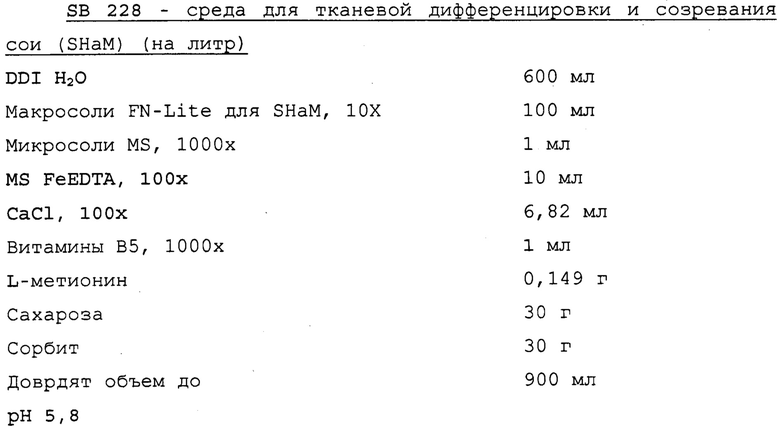
Автоклавируют
Добавляют в охлажденную среду (≤30°C):
*глутамин (конечная концентрация 30 нМ) 4% 110 мл
*Примечание: Конечный объем будет составлять 1010 мл после добавления глутамина.
Поскольку глутамин разлагается относительно быстро, его предпочтительно добавлять непосредственно перед применением среды. Истечение срока пригодности происходит через 2 недели после добавления глутамина; базовую среду можно хранить дольше без глутамина.

Доводят до объема
Автоклавируют

Доводят до объема
Автоклавируют

*EDTA должен полностью раствориться перед добавлением железа.
Доводят до объема
Раствор является светочувствительным. Флакон(ы) должны быть завернуты в фольгу во избежание света.
Автоклавируют

Доводят до объема
Автоклавируют
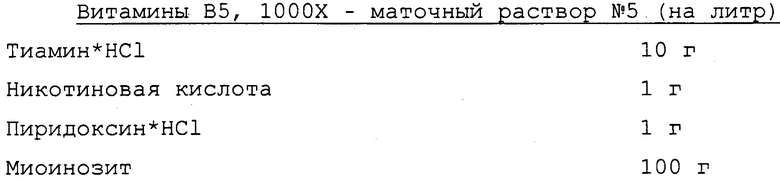
Доводят до объема
Хранят замороженным

Постепенно добавляют при перемешивании и применении слабого нагревания.
Не превышают температуру 35°C.
Доводят до объема
Стерилизуют фильтрованием
Хранят замороженным*
*Примечание: подогревают размороженный маточный раствор на бане температурой 31°C для полного растворения кристаллов.
Регенерация соматических зародышей сои в растения
Для получения целых растений из эмбриогенных суспензионных культур ткань нужно регенерировать. Обеспечивали созревание зародышей, как описано выше. После субкультивирования на среде SB103 в течение 3 недель отдельные зародыши можно удалить из кластеров и подвергнуть скринингу в отношении изменений в составе их жирных кислот, как описано в примере 7. Следует заметить, что на этой стадии можно подвергнуть скринингу любой выявляемый фенотип, являющийся результатом экспрессии генов, представляющих интерес. Он включает без ограничения изменения в профиле жирных кислот, профиль белков и их содержание, содержание углеводов, скорость роста, жизнеспособность или способность к нормальному развитию в растение сои.
Зрелые отдельные зародыши высушивали путем помещения их пустую небольшую чашку Петри (35×10 мм) примерно на 4-7 дней. Планшеты оборачивали волокнистой лентой (создавая небольшую камеру с регулируемой влажностью). Высушенные зародыши высаживали в среду SB71-4, где их оставили прорастать при тех же условиях культивирования, что описаны выше. Пророщенные саженцы удаляли из среды для прорастания и тщательно промывали водой, а затем высаживали в среду Redi-Earth в 24-ячеечный упаковочный поднос, накрывали прозрачным пластиковым куполом. Через 2 недели купол удаляли и растения подвергали закаливанию в течение дополнительной недели. Если саженцы выглядели закаленными, их пересаживали в 10" горшок со средой Redi-Earth в количестве до 3 саженцев на горшок. После 10-16 недель зрелые семена собирали, лущили и анализировали в отношении жирных кислот.
ПРИМЕР 16
Система для скрининга в отношении мегануклеазной активности на основе дрожжей
Создавали штаммы дрожжей для скрининга в качестве хозяев для скрининга в отношении мегануклеазной активности. Ген дрожжей Ade2 (Genetika 1987 Jul-23(7):1141-8) (SEQ ID NO: 36) использовали в качестве маркерного гена с видимым проявлением, а также в качестве селектируемого маркера на схеме, изображенной на фигуре 7. Фрагменты гена, соответствующие первым 1000 нуклеотидам кодирующей последовательности Ade2 (5'-фрагмент Ade2) и последним 1011 нуклеотидам кодирующей последовательности Ade2 (3'-фрагмент Ade2), разрушали с помощью
Между 5'-фрагментом Ade2 и 3'-фрагментом Ade2 находились 305 нуклеотидов дуплицированных последовательностей. Полученные в результате конструкции использовали для замещения гена Ade2 (положения нуклеотидов 566193-564480 в хромосоме 15) штамма дрожжей BY4247. Полученные в результате штаммы дрожжей для скрининга VER8145, VER8189 и HD1327 можно охарактеризовать как BY4742 MATa his3delta1 leu2delta0 lys2delta0 ura3delta0 Gal2+. Если разрезание мегануклеазой происходит между дуплицированными последовательностями, может происходить гомологичная рекомбинация, в результате чего образуется функциональный ген Ade2.
Образование функционального гена Ade2 можно применять в качестве критерия отбора: если дрожжевые клетки растут на среде с недостатком аденина, то к росту способны только те из них, которые имеют функциональный ген Ade2.
Образование функционального гена Ade2 также можно использовать в качестве критерия скрининга. Клетки дрожжей с функциональным геном Ade2 являлись белыми, тогда как таковые, у которых отсутствует функционирование Ade2, проявляли красную окраску в связи с более ранним накоплением метаболита в пути биосинтеза аденина, что в результате приводит к образованию красных колоний с белыми секторами, показанному на фигурах 8 и 9. Степень образования белых секторов, иногда распространяющегося на целые колонии, указывает на величину активности разрезания, присущей мегануклеазам. Поскольку фенотип образования секторов является качественным показателем мегануклеазной активности, задействовали числовую систему количественных показателей 0-4. Как показано на фигуре 9, количественный показатель 0 означает, что белые сектора не наблюдались (отсутствовало разрезание мегануклеазами); количественный показатель 4 означает полностью белые колонии (полное разрезание сайта распознавания); количественные показатели 1-3 означают промежуточные фенотипы образования белых секторов (и промежуточные степени разрезания сайта распознавания).
ПРИМЕР 17
Анализ вариантных последовательностей распознавания для мегануклеазы LIG3-4 и MHP14+ в маисе
Для демонстрации активности расщепления идентифицированных в данном документе вариантных сайтов распознавания in planta встречающийся в природе вариантный сайт распознавания (SEQ ID NO: 11) для мегануклеазы MHP14+ идентифицировали у маиса и активность его расщепления in planta (измеряемая по частоте мутагенеза сайта распознавания) и активность расщепления in vitro сравнивали с активностью расщепления предполагаемого сайта распознавания для MHP14+.
Для определения активности расщепления сайтов распознавания in planta плазмидную ДНК, содержащую кассету экспрессии для мегануклеазы MHP14+ доставляли в зародыши маиса посредством бомбардировки частицами для обеспечения возможности возникновения двунитевого разрыва с последующей ПЦР в реальном времени, которую осуществляли с анализами TaqMan, охватывающими сайты распознавания. Относительное число копий рассчитывали с помощью способа ΔΔCt относительно внутреннего контроля в анализе TaqMan с использованием нетрансформированных зародышей в качестве калибровочного стандарта. Зародыши с относительным числом копий менее 0,8 считали расщепленными и/или мутированными. Активность расщепления мегануклеазой in vitro анализировали, как описано в примере 9 на плазмидной ДНК.
Активность расщепления плазмидной ДНК in vitro в вариантном сайте распознавания SEQ ID NO:11 была примерно в 3 раза эффективнее, чем в предполагаемом сайте распознавания с SEQ ID NO: 14, в зависимости от температуры реакционной смеси (см. фигуру 10A). Подобную тенденцию наблюдали in planta, причем 35% зрелых демонстрировали расщепление в эндогенном вариантном сайте распознавания, что подтверждалось мутагенезом, хотя только 15% демонстрировали расщепление (мутагенез) в предполагаемом сайте распознавания (см. фигуру 10B).
Подобный анализ можно осуществлять для вариантных сайтов распознавания для LIG3-4, описанных в данном документе или для любых других идентифицированных вариантных сайтов распознавания. Ожидается, что частоты модификации сайта, в том числе делеции, вставки или любой комбинации этих двух модификаций, будут повышаться для других вариантных сайтов распознавания с улучшенной активностью расщепления, идентифицированных в примере 10 (-7C, 8T (Lig3-4); -11C, -7C, -2G, -1T, +8T (Lig3-4); -11C, -7C, -1T, +8T (Lig3-4); -3A, -2G, -1T, +2A, +7T, +8G, +11G (MHP14+); -3A, -2G, -1T, +2A, +7T, +8G, +11A (MHP14+); -3A, -2G, -1T, +7T, +8G, +11G (MHP14+); -2G, -1T, +2A, +7T, +8G, +11G (MHP14+); -2G, -1T, +7T, +8G, +11G (MHP14+), соответствующих SEQ ID NO: 13-35) или любого другого вариантного сайта распознавания, идентифицированного с помощью способа, описанного в данном документе, которые будут искусственно встраивать в геном.
ПРИМЕР 18
Анализ вариантных последовательностей распознавания для мегануклеазы LIG3-4 и MHP14+ у сои
Для тестирования предполагаемых и вариантных сайтов распознавания для мегануклеаз LIG3-4 и MHP14+ в двудольном растении, подобном сое, последовательности сайтов распознавания маиса можно клонировать в ДНК конструкции для трансформации и вводить в сою с помощью биолистической трансформации, как описано в примере 15.
Сравнение нескольких сайтов распознавания, расположенных в одном том же локусе
Для сравнения активности расщепления (эффективностей разрезания) у разных последовательностей распознавания можно расположить несколько последовательностей распознавания в одной ДНК конструкции и ввести разные конструкции, содержащие несколько сайтов распознавания предпочтительно в одном и том же геномном локусе для исключения эффектов расположения. Трансформационная система на основе FLP/FRT-опосредованной сайт-специфической интеграции является ценным инструментом для достижения вышеизложенной цели путем расположения разных донорных ДНК конструкций в ранее охарактеризованных целевых сайтах (Plant Physiology, Li et al., 2009; заявка на патент США №12/634775). После того, как сайты распознавания интегрировались в геном сои, трансгенные трансформанты с одной копией можно идентифицировать, характеризовать и выбирать в качестве новых материалов для последующей трансформации соответствующими мегануклеазами для оценки активности расщепления (эффективности разрезания) каждого сайта распознавания соответствующей ему мегануклеазой. Поскольку несколько сайтов распознавания введены в один и тот же геномный сайт, эффективность разрезания соответствующими мегануклеазами можно сравнить.
С этой целью предполагаемый сайт распознавания для LIG34 (SEQ ID NO:13), а также предполагаемые сайты распознавания для MHP14+ (SEQ ID NO:14) и MS26 (SEQ ID NO: 37) клонировали в донорную конструкцию для SSI PHP57712 (SEQ ID NO: 38) между геном селектируемого маркера GM-ALS и кассет с генами признаков сверхэкспрессии DGAT2 (диацилглицерол-ацилтрансферазы) для сверхэкспрессии для высокого содержания масла, искусственной микроРНК для косупрессии FAD3 (ω-3-десатуразы) для высокого содержания ненасыщенных жирных кислот и косупрессии GAS (галактинол-синтазы) с помощью шпильки для высокого уровня доступной энергии (фигура 11 A). Трансгенных трансформантов с донорной ДНК PHP57712, интегрированной в несколько ранее охарактеризованных геномных сайтах, получали с помощью биолистической SSI трансформации сои частицами, как описано выше. Трансгенных трансформантов с чистыми вставками генов признаков и последовательностей распознавания для мегануклеазы отбирали, и их будут использовать для следующего раунда трансформации мегануклеазами LIG34, MS26 и MHP14+ для исследования активности расщепления трех предполагаемых сайтов распознавания.
Сравнение предполагаемых сайтов распознавания по сравнению и вариантных сайтов распознавания, расположенных в одном и том же локусе
Вариантный сайт распознавания для LIG3-4 (SEQ ID NO:22) и вариантный сайт распознавания для MHP14+ (SEQ ID NO:35), а также предполагаемый сайт распознавания для MS26 (SEQ ID NO: 37) клонировали в другую донорную конструкцию SSI PHP62252 (SEQ ID NO: 39) (фигура 11 B) и трансформировали в некоторые из тех же геномных сайтов сои с помощью SSI трансформации. Трансгенных трансформантов с чистыми вставками генов признаков и последовательностей распознавания для мегануклеазы будут отбирать и будут использовать для другого раунда трансформации мегануклеазами MS26, MHP14+ и LIG34 для исследования активностей расщепления сайтов распознавания. Поскольку все сайты распознавания введены в одни и те же геномные сайты, эффективности разрезания различных сайтов распознавания можно сравнить более содержательно. Активность расщепления вариантных сайтов распознавания для LIG3-4 и MHP14+ (SEQ ID NO: 22 и 35) можно сравнить с активностью расщепления их предполагаемым сайтам распознавания (SEQ ID NO: 13 и 14), как описано в примере 17.
ПРИМЕР 19
Способ нацеливания вставки полинуклеотида, представляющего интерес, в специфический хромосомный сайт в геноме растения
Нуклеотидную последовательность, содержащую вариабельную последовательность распознавания средством для индукции двунитевого разрыва, вводили в геном целевого организма, достигая целевого сайта (содержащего вариабельную последовательность распознавания) для вставки нуклеотидных последовательностей, представляющих интерес. Можно затем получить библиотеку стабильных растений или культивируемых тканей, содержащую вариабельный сайт распознавания в различных положениях по всему геному растения.
Одним примером таких вариантных сайтов распознавания являются SEQ ID NO: 15, 16, 17, 18, 19, 20 и 21, которые могут расщепляться мегануклеазой LIG3-4, кодируемой SEQ ID NO: 1. Другим примером вариантных сайтов распознавания являются SEQ ID NO: 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34 и 35, которые могут расщепляться мегануклеазой MHP14+, кодируемой SEQ ID NO: 3. В одном варианте осуществления SEQ ID NO: 15, 16, 17, 18, 19, 20 и 21 и SEQ ID NO: 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34 и 35 не являются эндогенными в отношении генома маиса или растения.
После того, как были получены стабильное растение или культивируемая ткань, фрагмент ДНК, который содержит полинуклеотид, представляющий интерес, вводили в стабильно трансформированное растение или ткани в присутствии белка, индуцирующего двунитевой разрыв, такого как белок мегануклеазы. Этот процесс приводит в результате к вставке полинуклеотида, представляющий интерес, в вариабельную последовательность распознавания.
Считается, что трансформированное растение может содержать несколько целевых сайтов, например, без ограничения несколько сайтов распознавания, которые могут расщепляться средством для индукции двунитевого разрыва, а также сайты рекомбинации, такие как сайты FRT или сайты LOX. Примеры сайтов распознавания известны в данной области техники и включают сайты FRT (см., например, Schlake and Bode (1994) Biochemistry 33:12746-12751; Huang et al. (1991) Nucleic Acids Research 19:443-448).
ПРИМЕР 20
Улавливание геномных вариантных сайтов распознавания эндонуклеазой Cas и создание библиотек для глубокого секвенирования Illumina
Для улавливания в геномной ДНК вариантных сайтов распознавания для редкощепящих ДНК средств для индукции двунитевого разрыва, в которых большая часть расщепленных продуктов даст в результате тупоконечные концы, как например, для эндонуклеаз Cas (Gasiunas et al. (2012) Proc. Natl. Acad. Sci.USA 109:E2579-86, Jinek et al. (2012) Science 337:816-21), можно использовать присоединение аденина к 3'-концам расщепленного(ых) вариантного(ых) сайта(ов) распознавания в геномной ДНК. Адаптеры, содержащие комплементарный 3'-тиминовый липкий конец можно затем использовать для селективного лигирования с тупоконечными концами, образующимися в результате расщепления эндонуклеазой Cas, и обогащения ими.
Для создания материала для улавливания геномных вариантных сайтов распознавания будут проводить in vitro анализы расщепления и очистку будут проводить, по существу, как описано в примере 3, за исключением того, что вместо белка мегануклеазы будут использовать очищенный белок эндонуклеазы Cas и компонент(ы)-нуклеиновые кислоты, необходимые для образования функционального комплекса эндонуклеазы Cas, способного к расщеплению целевого сайта ДНК. Реакции in vitro можно проводить в разных буферах, при разных температурах или с разными значениями длины(длин) инкубации для получения идеальных условий расщепления эндонуклеазой Cas.
3'-Липкий конец из одного аденина будут затем присоединять к расщепленным эндонуклеазой Cas тупоконечным концам при инкубировании с расщепленной in vitro геномной ДНК при 37°C в течение 30 минут в 50 мкл реакционной смеси, содержащей ATP, 1× буфер Кленова (NEBnext) и 15 фрагмента Кленова (без экзонуклеазной активности) и очищать. Нефосфорилированные или фосфорилированные биотинилированные адаптеры, синтезированные и очищенные с помощью HPLC, содержащие 3'-тиминовый нуклеотидный липкий конец, комплементарный 3'-адениновому нуклеотидному липкому концу, можно затем лигировать с приблизительно 2 мкг расщепленной эндонуклеазой Cas удлиненной аденином 3' геномной ДНК в 100 мкл реакционной смеси с лигазой T4 (NEB). Полученные в результате лигированные с адаптером, расщепленные эндонуклеазой Cas предполагаемые и вариантные сайты распознавания можно затем обогащать, секвенировать и идентифицировать, подобно описанным в примерах 4, 6, 7 и 8.
| название | год | авторы | номер документа |
|---|---|---|---|
| БЫСТРЫЙ НАПРАВЛЕННЫЙ АНАЛИЗ СЕЛЬСКОХОЗЯЙСТВЕННЫХ КУЛЬТУР ДЛЯ ОПРЕДЕЛЕНИЯ ДОНОРНОЙ ВСТАВКИ | 2014 |
|
RU2668819C2 |
| МНОГОЦЕПОЧЕЧНЫЙ ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР И ЕГО ПРИМЕНЕНИЯ | 2013 |
|
RU2663725C2 |
| УНИВЕРСАЛЬНАЯ ДОНОРНАЯ СИСТЕМА ДЛЯ НАПРАВЛЕННОГО ВОЗДЕЙСТВИЯ НА ГЕНЫ | 2014 |
|
RU2687369C2 |
| СКОНСТРУИРОВАННАЯ СПОСОБАМИ ИНЖЕНЕРИИ ПЛАТФОРМА ДЛЯ ВСТРАИВАНИЯ ТРАНСГЕНА (ETIP) ДЛЯ НАЦЕЛИВАНИЯ ГЕНОВ И СТЭКИНГА ПРИЗНАКОВ | 2013 |
|
RU2666916C2 |
| НАПРАВЛЕННАЯ МОДИФИКАЦИЯ МАЛАТДЕГИДРОГЕНАЗЫ | 2013 |
|
RU2658437C2 |
| ОБОГАЩЕНИЕ АКТИВИРУЕМОЙ ФЛУОРЕСЦЕНЦИЕЙ СОРТИРОВКИ КЛЕТОК (FACS) ДЛЯ СОЗДАНИЯ РАСТЕНИЙ | 2013 |
|
RU2679510C2 |
| СПОСОБЫ ОБНАРУЖЕНИЯ ДНК ДЛЯ САЙТ-СПЕЦИФИЧЕСКОЙ НУКЛЕАЗНОЙ АКТИВНОСТИ | 2013 |
|
RU2678001C2 |
| ОПТИМАЛЬНЫЕ ЛОКУСЫ СОИ | 2014 |
|
RU2719150C2 |
| ЛОКУСЫ ФУНКЦИОНАЛЬНОСТИ FAD2 И СООТВЕТСТВУЮЩИЕ СПЕЦИФИЧНЫЕ К УЧАСТКУ-МИШЕНИ СВЯЗЫВАЮЩИЕ БЕЛКИ, СПОСОБНЫЕ ИНДУЦИРОВАТЬ НАПРАВЛЕННЫЕ РАЗРЫВЫ | 2013 |
|
RU2656159C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ЛЕЧЕНИЯ ГЕМОФИЛИИ В | 2011 |
|
RU2608643C2 |






























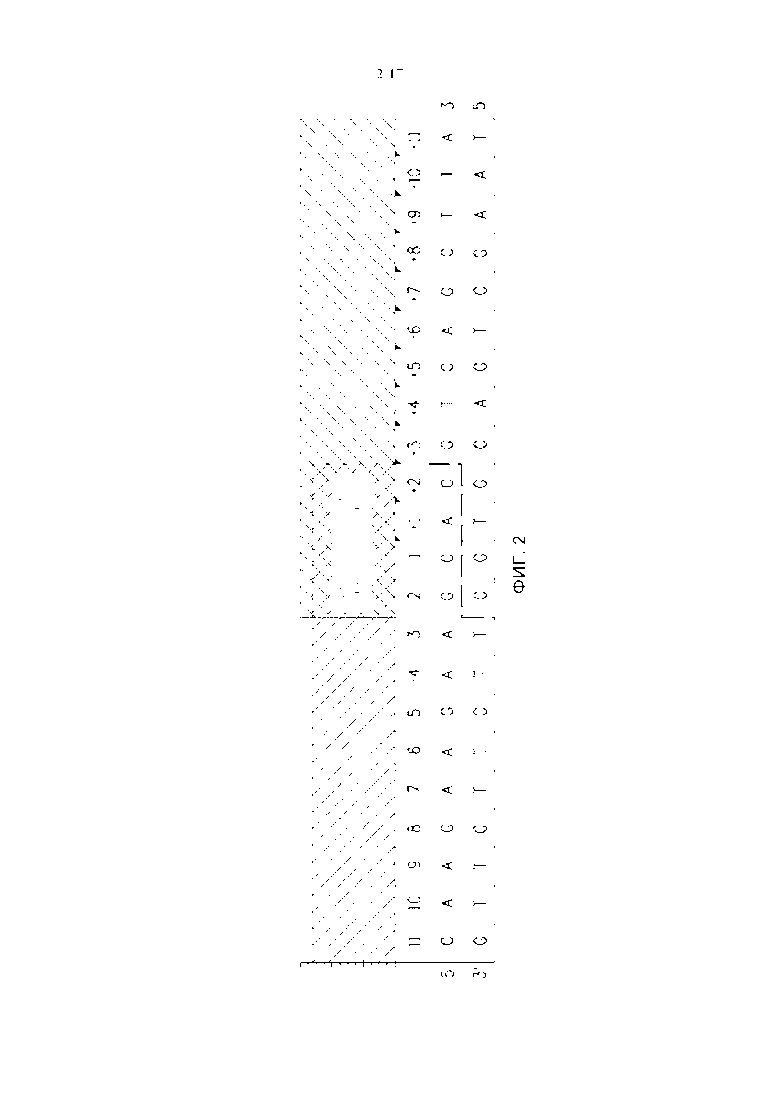

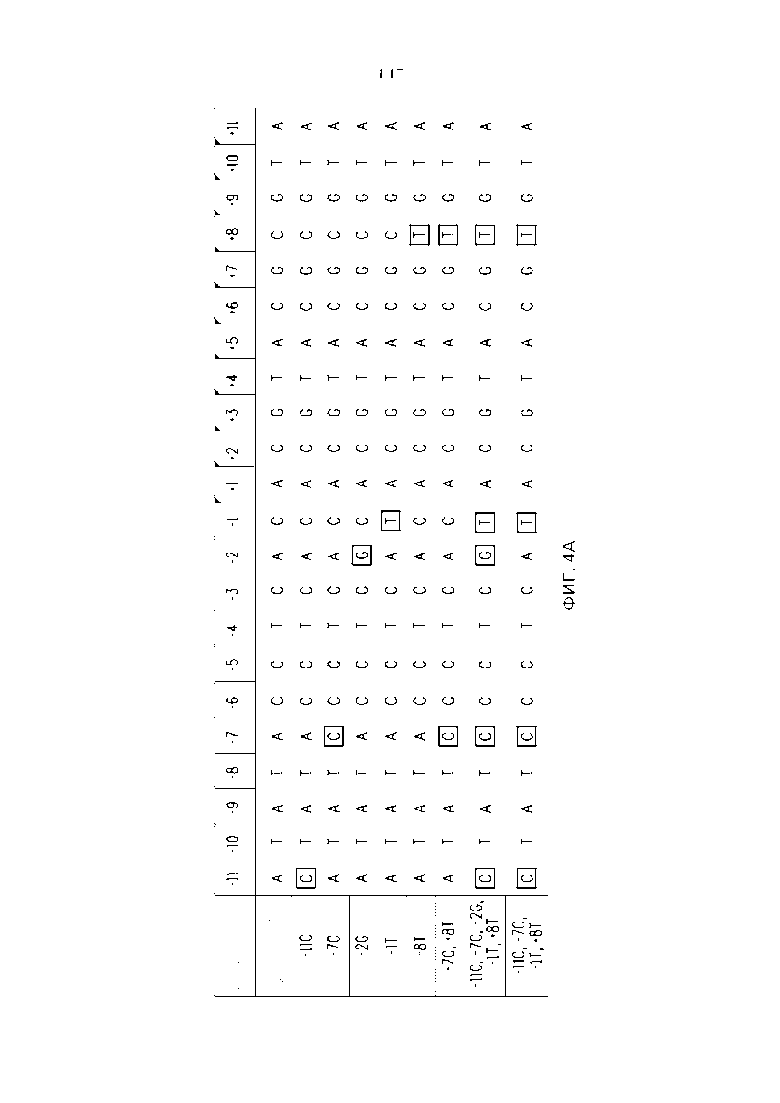

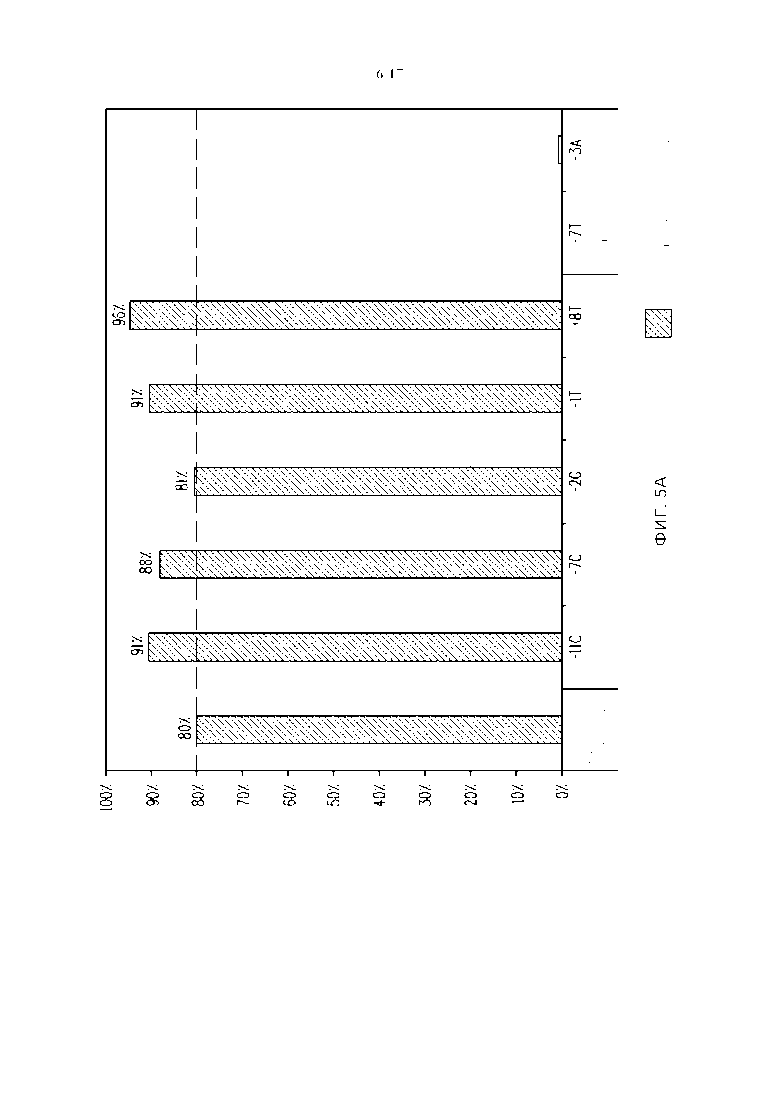
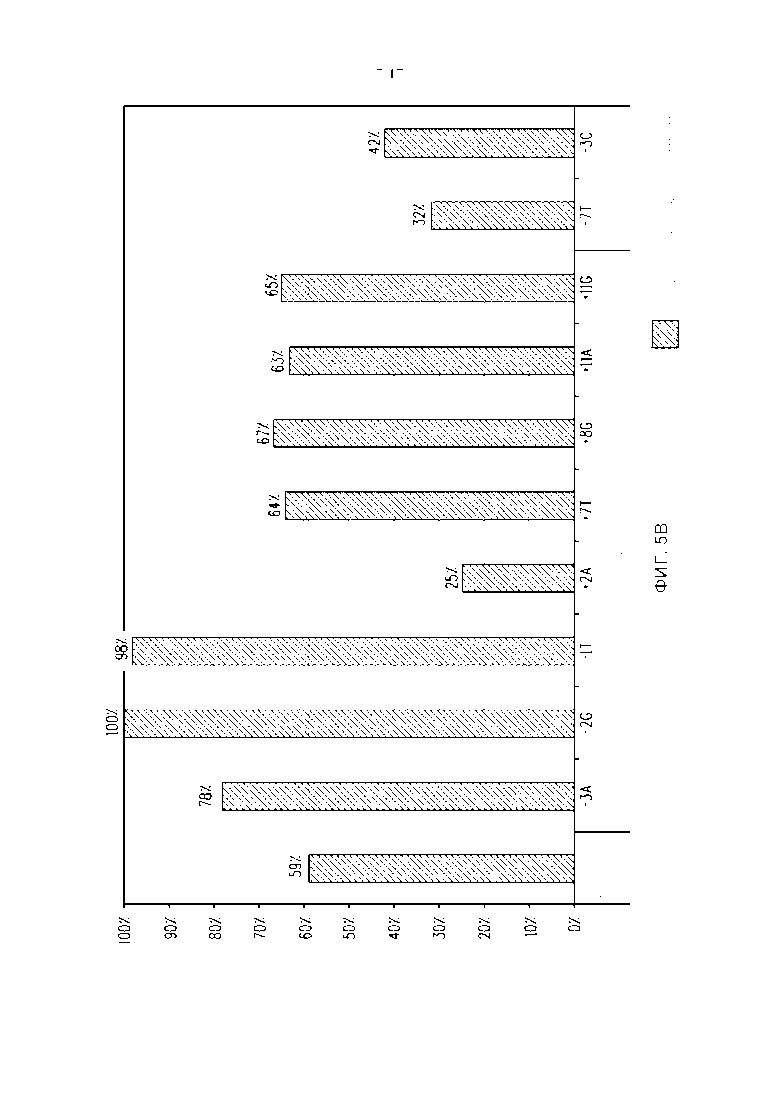
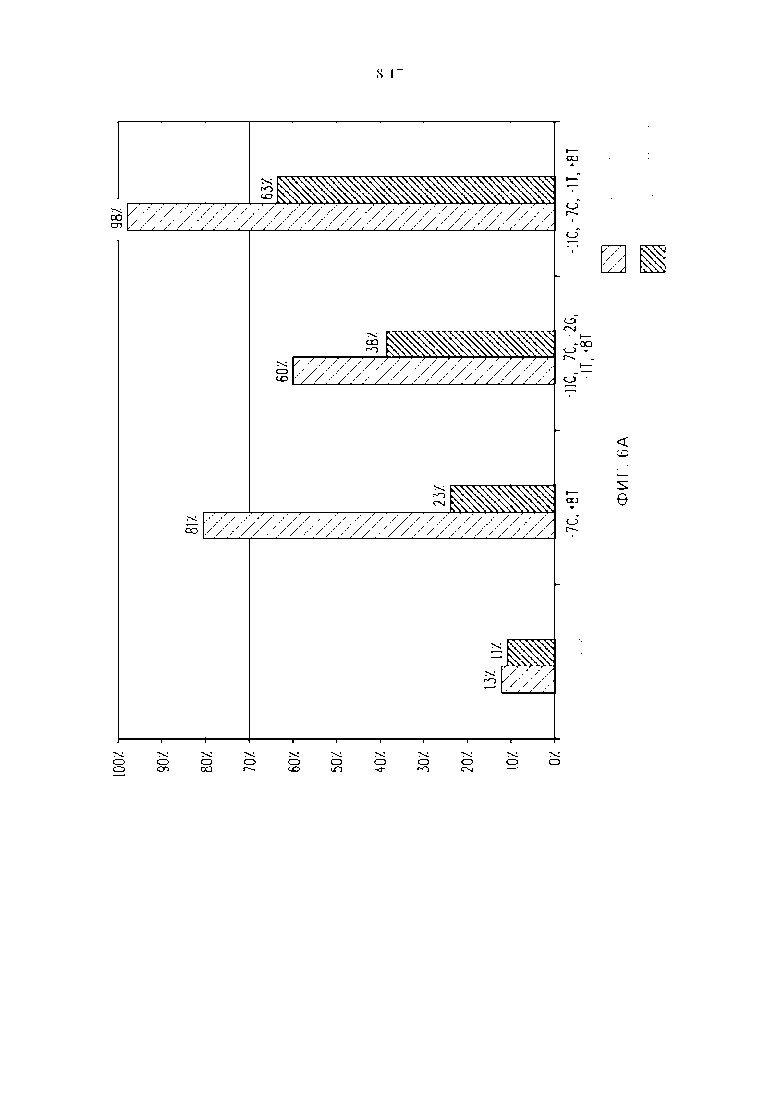
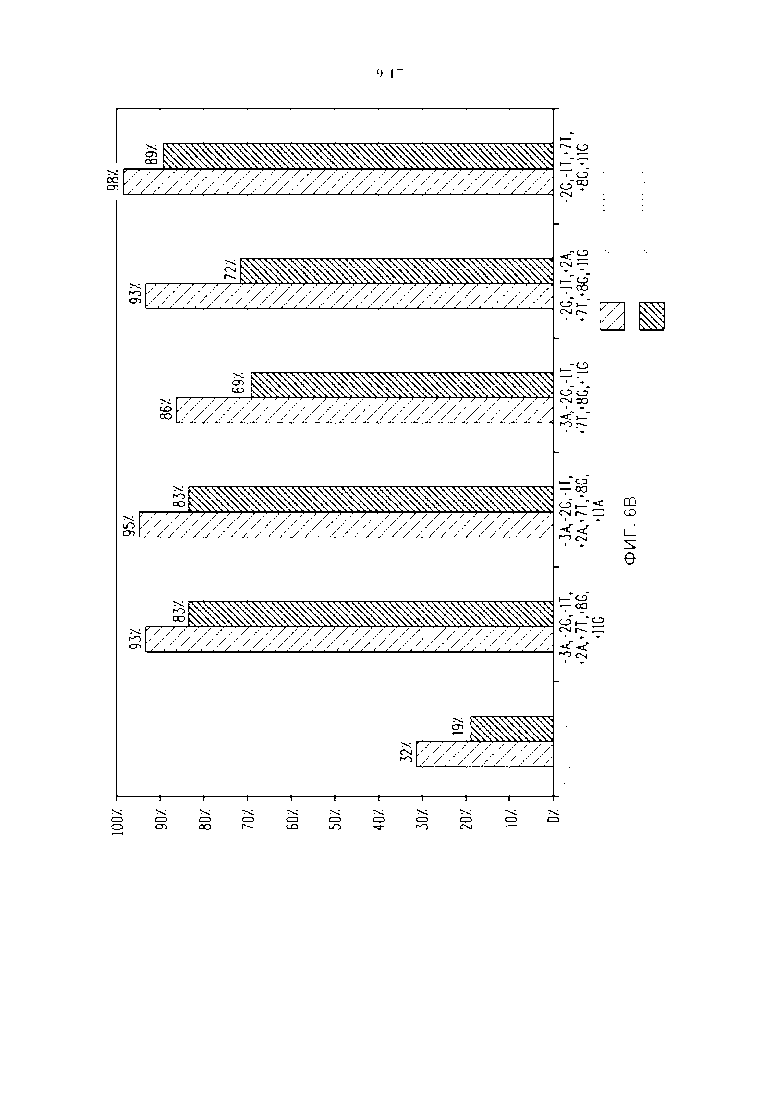

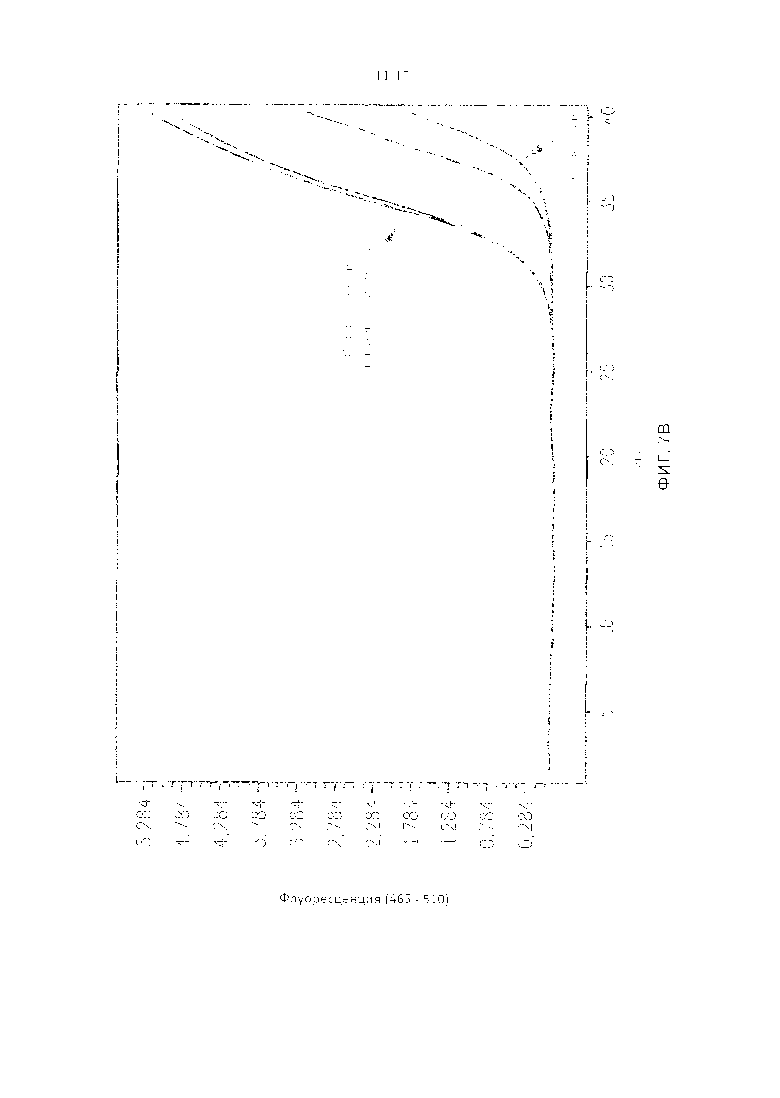
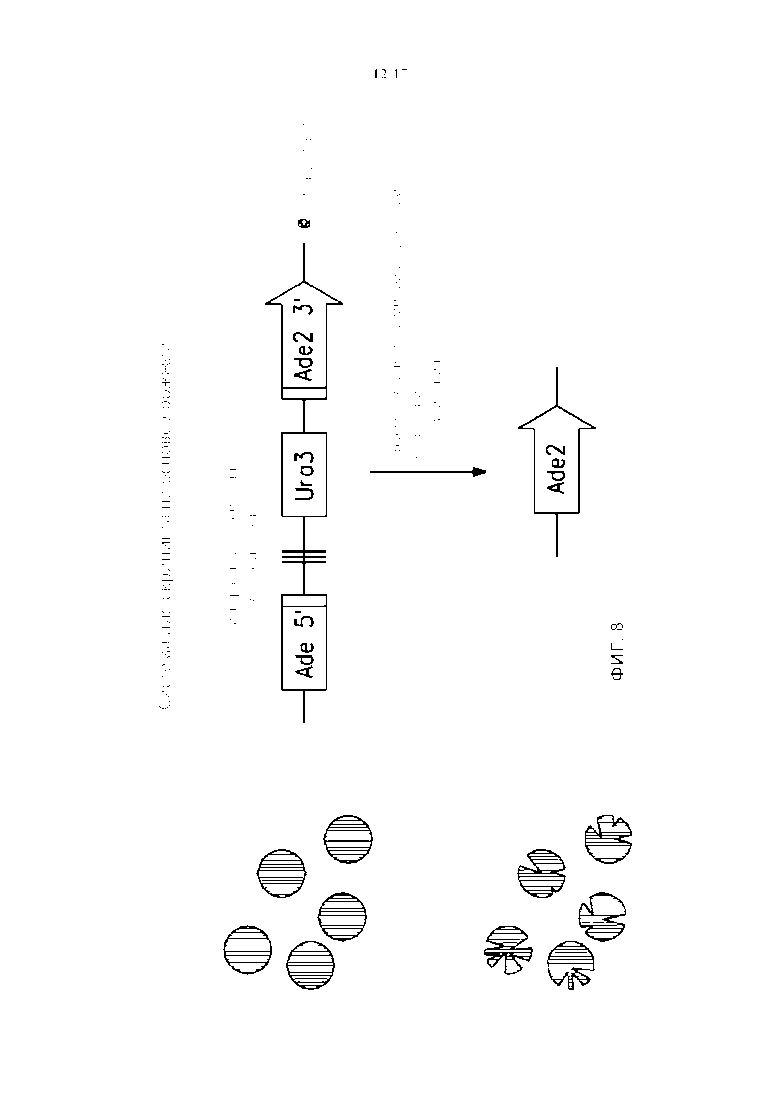
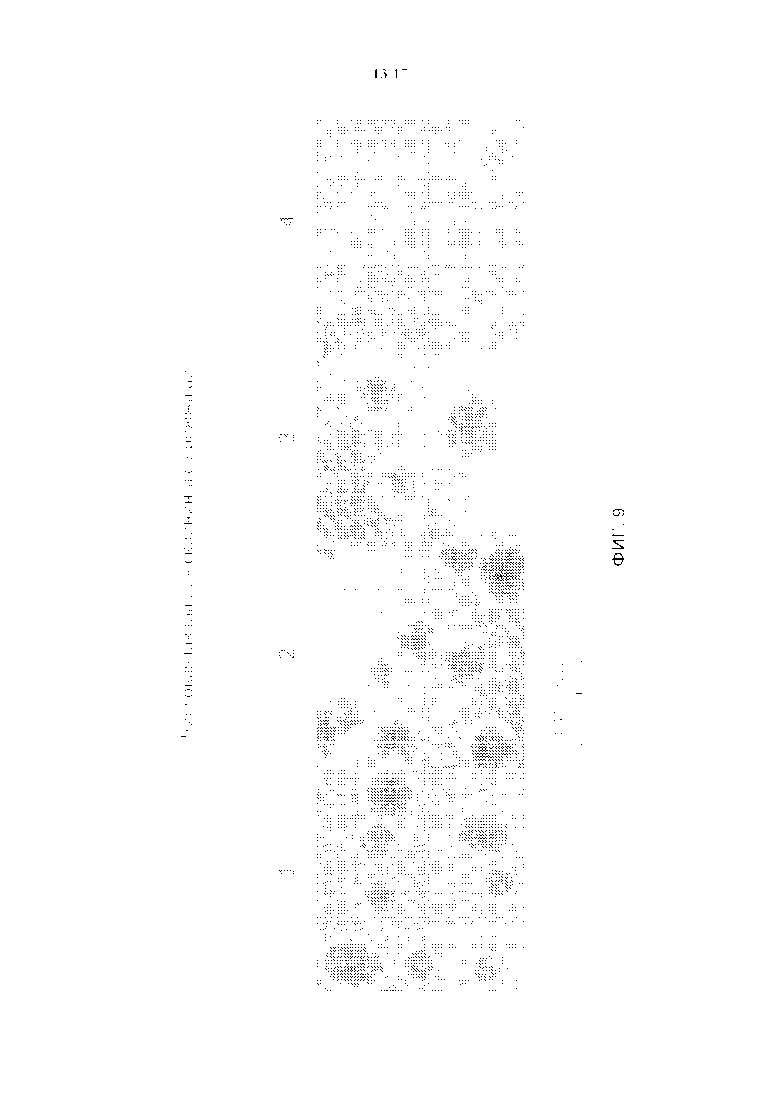
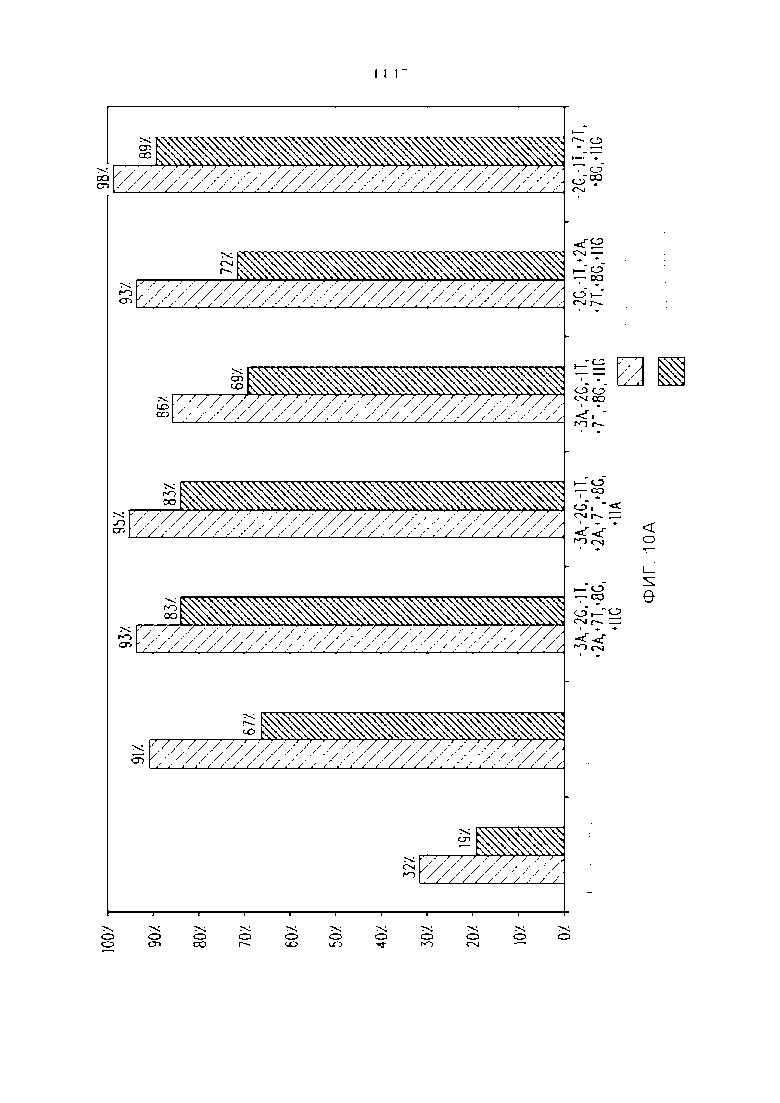
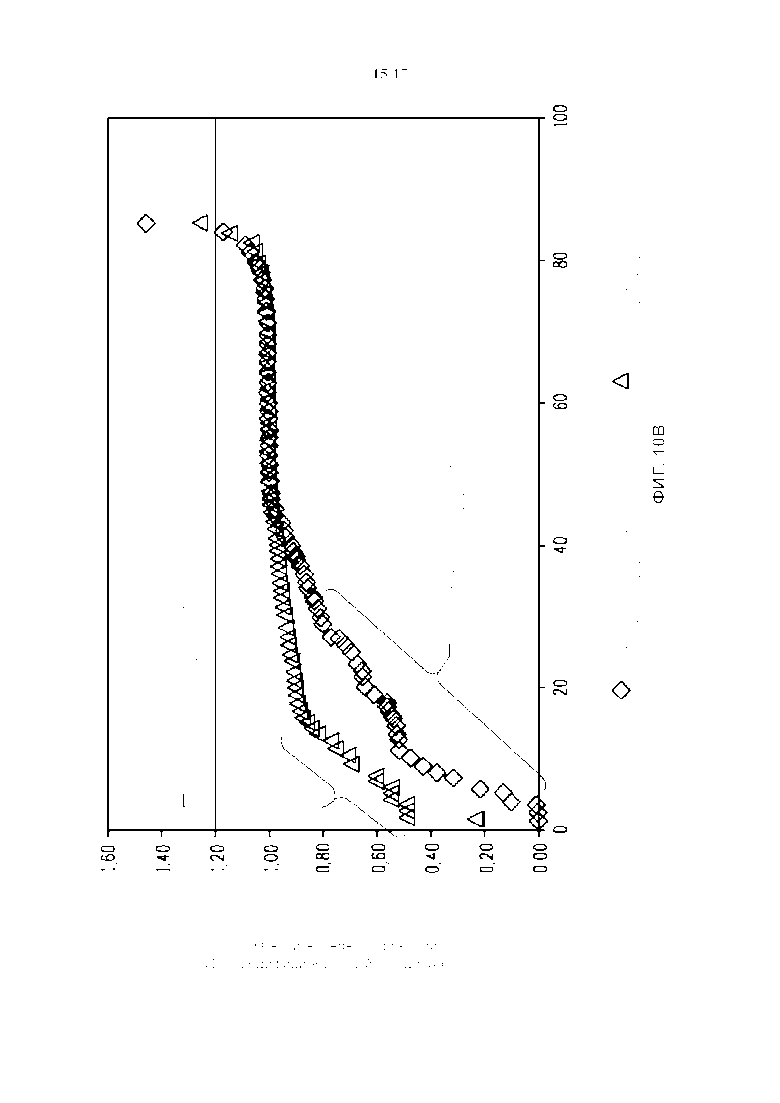
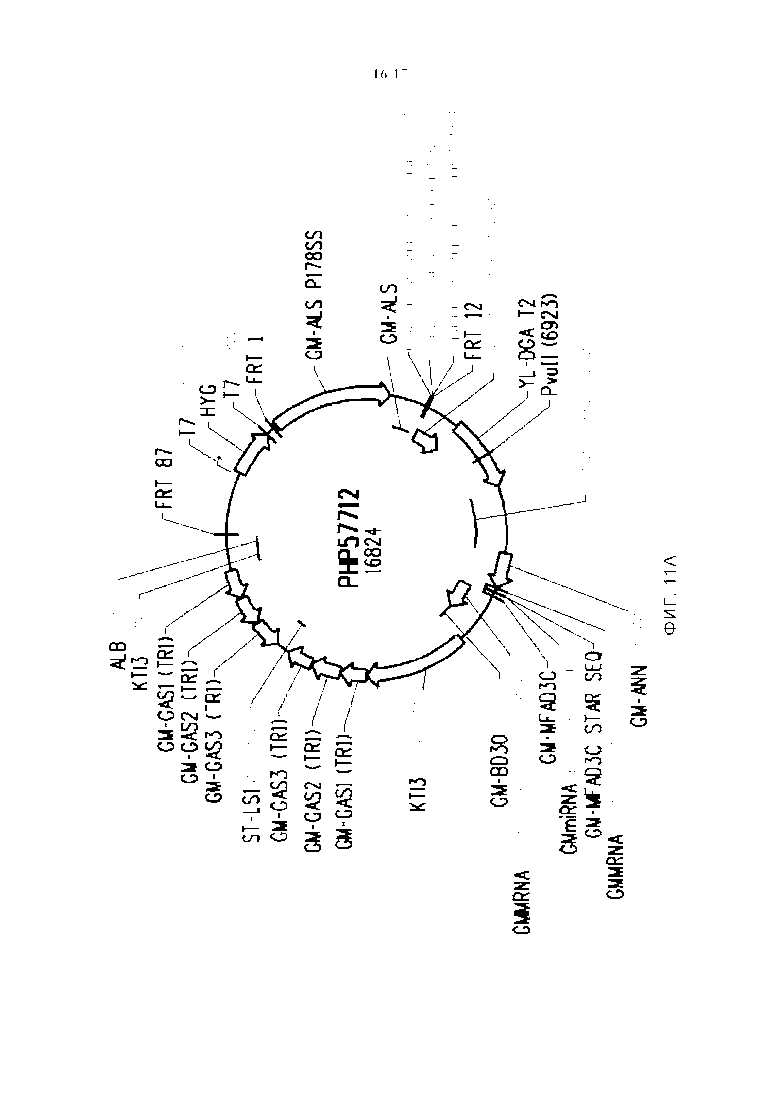
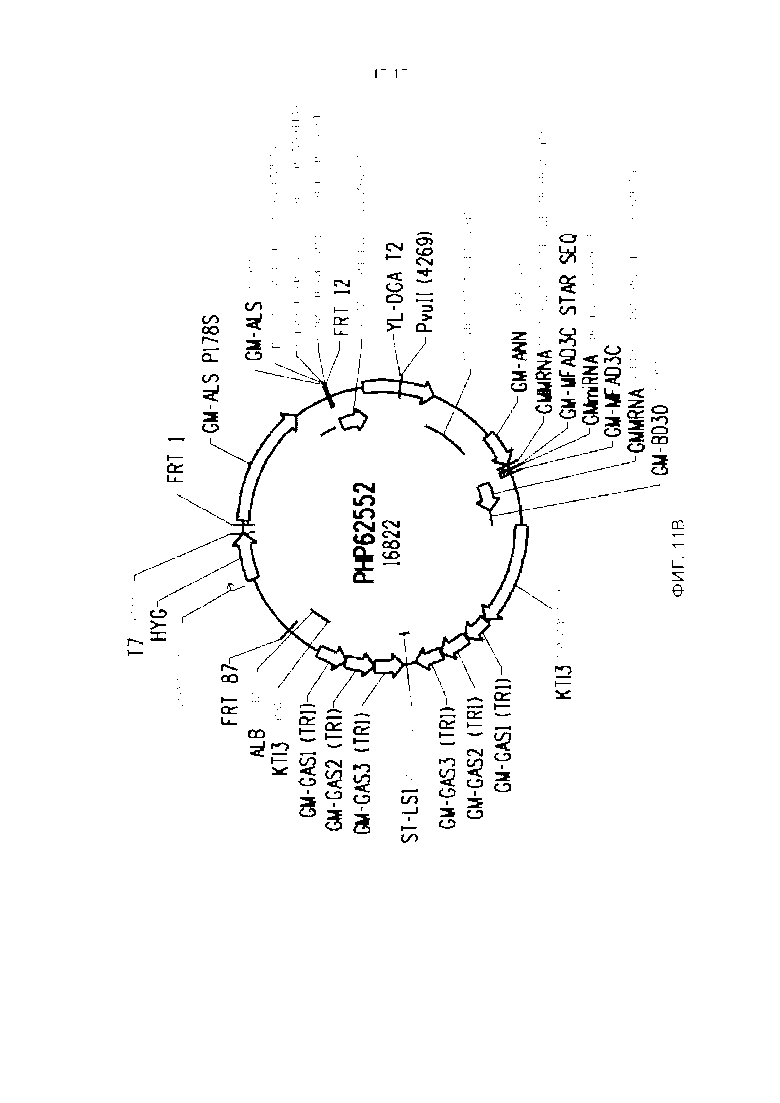
Изобретение относится к области биохимии, в частности к способам идентификации вариантного сайта распознавания для нуклеазы для индукции двунитевого разрыва. Изобретение позволяет эффективно осуществлять идентификацию вариантного сайта распознавания для нуклеазы для индукции двунитевого разрыва. 3 н. и 11 з.п. ф-лы, 11 ил., 3 табл., 20 пр.
1. Способ идентификации вариантного сайта распознавания для нуклеазы для индукции двунитевого разрыва, способной к введению редкого двунитевого разрыва в предполагаемом сайте распознавания, причем указанный способ включает:
a. приведение в контакт геномной ДНК с нуклеазой для индукции двунитевого разрыва, способным к введению двунитевого разрыва в указанную геномную ДНК, где двунитевой разрыв приводит в результате к образованию нуклеотидного липкого конца;
b. лигирование первого адаптера с указанным нуклеотидным липким концом;
c. разрезание лигированной ДНК, полученной на стадии (b), и лигирование по меньшей мере одного второго адаптера с разрезанным нуклеотидным концом для осуществления амплификации и секвенирования фрагментов геномной ДНК, окружающих двунитевой разрыв;
d. выравнивание нуклеотидных последовательностей фрагментов ДНК, полученных в (c), с эталонной геномной последовательностью ДНК; и
e. идентификацию вариантного сайта распознавания, содержащего изменение по меньшей мере по одному нуклеотидному основанию по сравнению с предполагаемым сайтом распознавания указанной сконструированной нуклеазы для индукции двунитевого разрыва.
2. Способ по п. 1, где нуклеазу для индукции двунитевого разрыва получают из I-CreI.
3. Способ идентификации вариантного сайта распознавания с улучшенной активностью расщепления для нуклеазы для индукции двунитевого разрыва, способной к введению редкого двунитевого разрыва в предполагаемом сайте распознавания, причем указанный способ включает:
a. приведение в контакт геномной ДНК с нуклеазой для индукции двунитевого разрыва, способным к введению двунитевого разрыва в указанную геномную ДНК, где двунитевой разрыв приводит в результате к образованию нуклеотидного липкого конца;
b. лигирование первого адаптера с указанным нуклеотидным липким концом;
c. разрезание лигированной ДНК, полученной на стадии (b), и лигирование второго адаптера с разрезанным нуклеотидным концом для осуществления амплификации и секвенирования фрагментов геномной ДНК, окружающих двунитевой разрыв;
d. выравнивание нуклеотидных последовательностей фрагментов ДНК, полученных в (c), с эталонной геномной последовательностью ДНК; и
e. идентификацию вариантного сайта распознавания, содержащего изменение по меньшей мере по одному нуклеотидному основанию по сравнению с предполагаемым сайтом распознавания указанной нуклеазы для индукции двунитевого разрыва;
f. анализ активности нуклеазы для индукции двунитевого разрыва в вариантных сайтах распознавания из d); и
g. идентификацию вариантного сайта распознавания, который приводит в результате к повышенной активности нуклеазы для индукции двунитевого разрыва по сравнению с активностью в предполагаемом сайте распознавания.
4. Способ по п. 3, где о повышенной активности нуклеазы для индукции двунитевого разрыва свидетельствует:
a. более высокий процент (%) расщепления вариантного сайта распознавания по сравнению с процентом (%) расщепления предполагаемого сайта распознавания, где сайты распознавания расположены на геномной ДНК;
b. более высокий процент (%) расщепления вариантного сайта распознавания по сравнению с процентом (%) расщепления предполагаемого сайта распознавания, где сайты распознавания расположены на плазмидной ДНК;
c. более высокая оценка в анализе на дрожжах для вариантного сайта распознавания по сравнению с предполагаемым сайтом распознавания; или
d. любая комбинация (a), (b) и (c).
5. Способ идентификации вариантного сайта распознавания для нуклеазы для индукции двунитевого разрыва, способной к введению редкого двунитевого разрыва в предполагаемом сайте распознавания, причем указанный способ включает:
a. приведение в контакт геномной ДНК с нуклеазой для индукции двунитевого разрыва, способной к введению двунитевого разрыва в указанную геномную ДНК, где двунитевой разрыв приводит в результате к образованию тупого конца;
b. создание нуклеотидного липкого конца из тупого конца из (a);
c. лигирование первого адаптера с нуклеотидным липким концом из (b);
d. разрезание лигированной ДНК, полученной на стадии (c), и лигирование по меньшей мере одного второго адаптера с разрезанным нуклеотидным концом для осуществления амплификации и секвенирования фрагментов геномной ДНК, окружающих двунитевой разрыв;
e. выравнивание нуклеотидных последовательностей фрагментов ДНК, полученных в (d), с эталонной геномной последовательностью ДНК; и
f. идентификацию вариантного сайта распознавания, содержащего изменение по меньшей мере по одному нуклеотидному основанию по сравнению с предполагаемым сайтом распознавания указанной сконструированной нуклеазы для индукции двунитевого разрыва.
6. Способ по п. 5, где нуклеаза для индукции двунитевого разрыва представляет собой эндонуклеазу Cas.
7. Способ по п. 6, где эндонуклеаза Cas способна к образованию комплекса с направляющей РНК, где указанный комплекс позволяет эндонуклеазе Cas вводить двунитевой разрыв в указанную геномную ДНК.
8. Способ по п. 1, 3 или 5, где нуклеазу для индукции двунитевого разрыва выбирают из группы, состоящей из мегануклеазы, нуклеазы "цинковые пальцы", TAL-эффекторной нуклеазы, транспозазы, эндонуклеазы Cas и сайт-специфической рекомбиназы.
9. Способ по п. 1, 3 или 5, где нуклеотидный липкий конец представляет собой 3'-нуклеотидный липкий конец.
10. Способ по п. 1, 3 или 5, где нуклеотидный липкий конец представляет собой 5'-нуклеотидный липкий конец.
11. Способ по п. 1, 3 или 5, где первый адаптер, лигированный с нуклеотидным липким концом, представляет собой нефосфорилированный по 5' адаптер.
12. Способ по п. 1, 3 или 5, где геномную ДНК выбирают из группы, состоящей из прокариотической ДНК, эукариотической ДНК и синтетической ДНК.
13. Способ по п. 12, где эукариотическую ДНК выделяют из растения, дрожжей или животного.
14. Способ по п. 13, где растение выбирают из группы, состоящей из сои, подсолнечника, хлопчатника, люцерны, канолы, табака, картофеля, Arabidopsis, сафлора, маиса, риса, сорго, ячменя, пшеницы, проса, овса, сахарного тростника, газонной травы и проса прутьевидного.
| WO 2013006745 A2, 10.01.2013 | |||
| WO 2009006297 A2, 08.01.2009 | |||
| WO 2012129373 A2, 27.09.2012 | |||
| US 2011165679 A1, 07.07.2011 | |||
| ЛУТОВА Л | |||
| А., Биотехнология высших растений, Издательство С.-Петербургского университета, 2010, стр | |||
| Способ крашения тканей | 1922 |
|
SU62A1 |
Авторы
Даты
2019-07-16—Публикация
2014-03-10—Подача