ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННЫЕ ЗАЯВКИ
Настоящая заявка испрашивает приоритет в соответствии с § 119(e) раздела 35 U.S.C. предварительной заявки на патент США 61/899,566, поданной 4 ноября 2013 года, и предварительной заявки на патент США 61/889,587, поданной 4 ноября 2013 года, содержание которых полностью включено в настоящую заявку посредством отсылки.
ССЫЛКА НА СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ, ПРЕДСТАВЛЕННЫЙ В ЭЛЕКТРОННОЙ ФОРМЕ
Официальная копия списка последовательностей представлена в электронной форме через систему EFS-Web в виде списка последовательностей в формате ASCII в файле под названием "74892232308seqlist.txt", созданном 3 ноября 2014 года и имеющем размер 13,4 мегабайта, и подана одновременно с описанием. Список последовательностей, содержащийся в данном документе в формате ASCII, является частью описания и полностью включен в настоящую заявку посредством отсылки.
ССЫЛКА НА СПИСОК ТАБЛИЦ, ПРЕДСТАВЛЕННЫЙ В ЭЛЕКТРОННОЙ ФОРМЕ
Официальная копия списка таблиц представлена в электронной форме через систему EFS-Web в виде списка таблиц в формате PDF в файле под названием "Table3", созданном 03 ноября 2014 года и имеющем размер 11,6 мегабайта, и подана одновременно с описанием. Список таблиц, содержащийся в данном документе в формате PDF, является частью описания и полностью включен в настоящую заявку посредством отсылки.
УРОВЕНЬ ТЕХНИКИ
Геном многих типов двудольных растений, например растений сои, успешно трансформировали трансгенами в начале 1990-х годов. За последние двадцать лет было разработано множество методик трансформации генома двудольных растений, таких как соя, в которых трансген стабильно интегрируется в геном двудольных растений. Такое развитие методик трансформации двудольных обеспечило возможность успешно вводить трансген, включающий агрономический признак, в геном двудольных растений, таких как соя. Введение признаков устойчивости к насекомым и невосприимчивости к гербицидам в двудольные растения в конце 1990-х годов дало производителям новую и удобную технологическую инновацию для борьбы с насекомыми и широким спектром сорных трав, которая была беспрецедентной в разработке методов сельского хозяйства. В настоящее время трансгенные двудольные растения коммерчески доступны во многих странах, при этом новые трансгенные продукты, такие как соя EnlistTM, предлагают усовершенствованные решения для постоянно растущих вызовов в борьбе с сорняками. Применение трансгенных двудольных растений в современной агротехнике было бы невозможно, если бы не создание и совершенствование методик трансформации.
Впрочем, современные методики трансформации основаны на случайной вставке трансгенов в геном двудольных растений, таких как соя. Надежность случайной вставки генов в геном имеет несколько ограничений. Трансгенные события могут случайно интегрироваться в транскрипционные последовательности генов, прерывая таким образом экспрессию эндогенных признаков и изменяя рост и развитие растения. Кроме того, трансгенные события могут беспорядочно интегрироваться в участки генома, которые чувствительны к сайленсингу генов, что в итоге приводит к снижению или полному ингибированию экспрессии трансгена в первом или последующих поколениях трансгенных растений. Наконец, случайная интеграция трансгенов в геном растения требует значительных усилий и затрат при идентификации положения трансгенного события и отборе трансгенных объектов, которые функционируют, как предполагалось изначально, без агрономического воздействия на растение. Требуется непрерывно разрабатывать новые анализы для определения точного положения интегрированного трансгена в каждом трансгенном объекте, таком как трансгенная соя. Случайная природа методик трансформации растений приводит к "эффекту положения" интегрированного трансгена, который снижает эффективность методик трансформации.
Направленная модификация генома растений являлась давней и недостижимой целью как прикладных, так и фундаментальных исследований. Направленное введение генов и пакетов генов в определенные положения в геноме двудольных растений, таких как растения сои, улучшает качество трансгенных объектов, снижает затраты, связанные с получением трансгенных объектов, и обеспечивает новые способы создания трансгенных растительных продуктов, такие как последовательное пакетирование генов. В целом, направление трансгенов в определенные участки генома, вероятно, будет коммерчески выгодным. Значительные успехи были достигнуты в последние несколько лет в разработке способов и композиций для направленного воздействия и расщепления геномной ДНК с помощью сайт-специфических нуклеаз (например, цинк-пальцевых нуклеаз (ZFN), мегануклеаз, нуклеаз TALENS (от англ. transcription activator-like effector nucelases - подобные активаторам транскрипции эффекторные нуклеазы) и CRISPR-ассоциированной (от англ. clustered regularly interspaced short palindromic repeats - кластерные, разделенные регулярными интервалами, короткие палиндромные повторы) нуклеазы (CRISPR/Cas) со сконструированной crРНК/tracrРНК) для индукции направленного мутагенеза, индукции направленных делеций клеточных последовательностей ДНК и обеспечения направленной рекомбинации экзогенного донорного ДНК полинуклеотида в заданный геномный локус. См., например, патентные публикации США 20030232410; 20050208489; 20050026157; 20050064474 и 20060188987, и Международную патентную публикацию WO 2007/014275, описания которых полностью включены посредством отсылки во всех отношениях. В патентной публикации США 20080182332 описано применение неканонических цинк-пальцевых нуклеаз (ZFN) для направленной модификации геномов растений и в патентной публикации США 20090205083 описана ZFN-опосредованная направленная модификация геномного локуса EPSPs растений. Существующие способы направленной вставки экзогенной ДНК обычно включают котрансформацию растительной ткани донорным ДНК полинуклеотидом, содержащим по меньшей мере один трансген и сайт-специфическую нуклеазу (например, ZFN), которая создана для связывания и расщепления определенного геномного локуса активно транскрибируемой кодирующей последовательности. Это приводит к стабильной вставке донорного ДНК полинуклеотида в расщепленный геномный локус с направленным введением гена в указанный геномный локус, включающий активно транскрибируемую кодирующую последовательность.
Альтернативный вариант состоит в направлении трансгена в предварительно выбранные целевые негенные локусы в геноме таких двудольных растений, как соя. В последние годы несколько технологий разработали и применили к растительным клеткам для направленной доставки трансгена в геном двудольных растений, таких как соя. Впрочем, намного меньше известно о признаках геномных участков, которые подходят для таргетинга. Исторически в качестве локусов для таргетинга использовались несущественные гены и участки интеграции патогенов (вирусов) в геномах. Количество таких участков в геномах скорее является лимитирующим, и поэтому существует потребность в идентификации и исследовании оптимальных геномных локусов-мишеней, которые могут использоваться для таргетинга донорных полинуклеотидных последовательностей. В дополнение к возможности таргетинга, оптимальные геномные локусы, как ожидают, будут нейтральными участками, которые смогут поддерживать экспрессию трансгена и применение в селекции. Существует потребность в композициях и способах, которые определяют критерии для идентификации оптимальных негенных локусов в геноме двудольных растений, например растений сои, для направленной интеграции трансгена.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Один вариант осуществления настоящего описания направлен на способы идентификации оптимальных участков в геноме двудольного растения, включая, например, геном сои, для вставки экзогенных последовательностей. В литературе имеются сведения, которые предполагают, что хромосомные области растения пригодны для таргетинга и поддерживают экспрессию. Заявители создали набор критериев для идентификации областей нативных геномных последовательностей сои, которые являются оптимальными участками для сайт-направленной вставки. Более конкретно, в соответствии с одним вариантом осуществления, оптимальный локус должен быть негенным, поддерживать таргетинг и экспрессию гена, являться агрономически нейтральным и иметь подтверждение рекомбинации. Как раскрыто в настоящей заявке, заявители обнаружили множество локусов в геноме сои, которые соответствуют указанным критериям и, таким образом, представляют оптимальные участки для вставки экзогенных последовательностей.
В соответствии с одним вариантом осуществления в настоящей заявке раскрыта рекомбинантная последовательность сои, где рекомбинантная последовательность включает негенную геномную последовательность сои длиной по меньшей мере 1 тпн и целевую ДНК, вставленную в негенную геномную последовательность сои, где негенная геномная последовательность сои была изменена в результате вставки целевой ДНК. В одном варианте осуществления нативная негенная последовательность сои является гипометилированной, экспрессируемой, представляет подтверждение рекомбинации и расположена в проксимальном положении относительно генной области в геноме сои. В одном варианте осуществления негенная последовательность имеет длину в пределах от приблизительно 1 тпн до приблизительно 5,7 тпн. В одном варианте осуществления целевая ДНК включает экзогенные последовательности ДНК, включающие, например, регуляторные последовательности, сайты рестрикции, РНК-кодирующие области или белок-кодирующие области. В одном варианте осуществления целевая ДНК включает кассету экспрессии гена, включающую один или более трансгенов.
В соответствии с одним вариантом осуществления предложена рекомбинантная последовательность, включающая оптимальную негенную геномную последовательность сои длиной от приблизительно 1 тпн до приблизительно 5,7 тпн и целевую ДНК, где негенная геномная последовательность сои обладает 1, 2, 3, 4 или 5 из следующих свойств или особенностей:
a) имеет известную или предсказанную кодирующую последовательность сои в 40 тпн указанной геномной последовательности сои;
b) имеет последовательность, включающую 2 тпн до и/или 1 тпн после известного гена сои в 40 тпн от одного конца указанной геномной последовательности сои;
c) не содержит больше 1% метилирования ДНК в последовательности;
d) не содержит 1 тпн последовательность, обладающую более чем 40% идентичностью последовательности с любой другой последовательностью в геноме сои; и
e) представляет подтверждение рекомбинации с частотой рекомбинации больше 0,01574 сМ/мпн.
В соответствии с одним вариантом осуществления предложены растение сои, часть растения сои или клетка растения сои, включающие целевую ДНК, вставленную в идентифицированную и являющуюся мишенью негенную геномную последовательность сои в растении сои, части растения сои или клетке растения сои. В одном варианте осуществления негенная геномная последовательность сои в растении сои, части растения сои или клетке растения сои является гипометилированной, экспрессируемой, представляет подтверждение рекомбинации и расположена в проксимальном положении относительно генной области в геноме сои. В одном варианте осуществления негенная геномная последовательность сои в растении сои, части растения сои или клетке растения сои имеет длину от приблизительно 1 тпн до приблизительно 5,7 тпн, является гипометилированной и обладает 1, 2, 3 или 4 из следующих свойств или особенностей:
a) имеет известную или предсказанную кодирующую последовательность сои в 40 тпн указанной геномной последовательности сои;
b) имеет последовательность, включающую 2 тпн до и/или 1 тпн после известного гена сои в 40 тпн от одного конца указанной геномной последовательности сои;
c) не содержит больше 1% метилирования ДНК в последовательности;
d) не включает 1 тпн последовательность, обладающую более чем 40% идентичностью последовательности с любой другой последовательностью в геноме сои; и
e) представляет подтверждение рекомбинации с частотой рекомбинации больше 0,01574 сМ/мпн.
В одном варианте осуществления предложен способ создания трансгенной растительной клетки, включающей целевую ДНК, направленную в негенную геномную последовательность сои, где способ включает:
a) выбор оптимального негенного геномного локуса сои;
b) введение сайт-специфической нуклеазы в растительную клетку, где сайт-специфическая нуклеаза расщепляет указанную негенную последовательность;
c) введение целевой ДНК в растительную клетку;
d) направление целевой ДНК в указанную негенную последовательность, где расщепление указанной негенной последовательности вызывает интеграцию полинуклеотидной последовательности в указанную негенную последовательность; и
e) отбор трансгенных растительных клеток, включающих целевую ДНК, направленную в указанную негенную последовательность.
В соответствии с одним вариантом осуществления выбранная негенная последовательность включает 2, 3, 4, 5, 6, 7 или 8 из следующих особенностей:
a) негенная последовательность не содержит метилированный полинуклеотид;
b) негенная последовательность демонстрирует частоту рекомбинации от 0,01574 до 83,52 сМ/мпн в геноме сои;
c) негенная последовательность демонстрирует уровень занятости нуклеосомами генома сои от 0 до 0,494;
d) негенная последовательность обладает менее чем 40% идентичностью последовательности с любой другой 1 тпн последовательностью, содержащейся в геноме сои;
e) негенная последовательность имеет относительное значение местоположения от 0 до 0,99682 отношения геномного расстояния от центромеры хромосомы сои;
f) негенная последовательность имеет процентное содержание гуанина/цитозина в диапазоне от 14,36 до 45,9%;
g) негенная последовательность расположена проксимально к генной последовательности; и
h) 1 мпн область геномной последовательности сои, включающая указанную негенную последовательность, включает одну или более негенных последовательностей.
Вариант осуществления настоящего описания направлен на способы идентификации негенной геномной последовательности сои, включающие следующие этапы:
a) идентификация геномных последовательностей сои длиной по меньшей мере 1 тпн, которые не содержат более чем 1% уровень метилирования, с получением первого пула последовательностей;
b) исключение любых геномных последовательностей сои, которые кодируют транскрипты сои, из первого пула последовательностей;
c) исключение любых геномных последовательностей сои, которые не обеспечивают подтверждение рекомбинации, из первого пула последовательностей;
d) исключение любых геномных последовательностей сои, которые включают 1 тпн последовательность, которая обладает 40% или более высокой идентичностью последовательности с другой 1 тпн последовательностью, содержащейся в геноме сои, из первого пула последовательностей;
e) исключение любых геномных последовательностей сои, которые не имеют известного гена сои в 40 тпн идентифицированной последовательности, из первого пула последовательностей; и
f) идентификация оставшихся геномных последовательностей сои в пуле последовательностей как негенные геномные последовательности сои. После идентификации последовательностей их могут подвергать манипуляциям с использованием генно-инженерных методик для направления вставки последовательностей нуклеиновых кислот, не обнаруженных в локусах в нативном геноме.
В соответствии с вариантом осуществления любые геномные последовательности сои, которые не имеют известного гена сои, или по меньшей мере 2 тпн последовательности до или 1 тпн последовательности после известного гена, расположенной в пределах 40 тпн геномной последовательности сои, исключаются из пула негенных геномных последовательностей сои.
В соответствии с вариантом осуществления любые геномные последовательности сои, которые не имеют гена, экспрессирующего белок сои, расположенного в пределах 40 тпн геномной последовательности сои, исключаются из пула негенных геномных последовательностей сои.
В соответствии с одним вариантом осуществления в настоящей заявке раскрыта очищенная полинуклеотидная последовательность сои, где очищенная последовательность включает негенную геномную последовательность сои длиной по меньшей мере 1 тпн. В одном варианте осуществления негенная последовательность сои является гипометилированной, экспрессируемой, представляет подтверждение рекомбинации и расположена в проксимальном положении относительно генной области в геноме сои. В одном варианте осуществления негенная последовательность имеет длину в пределах от приблизительно 1 тпн до приблизительно 5,7 тпн. В одном варианте осуществления целевая ДНК включает экзогенные последовательности ДНК, включающие, например, регуляторные последовательности, сайты рестрикции, РНК-кодирующие области или белок-кодирующие области. В одном варианте осуществления целевая ДНК включает кассету экспрессии гена, включающую один или более трансгенов.
В соответствии с одним вариантом осуществления предложена очищенная полинуклеотидная последовательность сои, включающая оптимальную негенную геномную последовательность сои длиной от приблизительно 1 тпн до приблизительно 5,7 тпн и целевую ДНК, где негенная геномная последовательность сои обладает 1, 2, 3, 4 или 5 из следующих свойств или особенностей:
a) имеет известную или предсказанную кодирующую последовательность сои в пределах 40 тпн указанной рекомбинантной последовательности;
b) имеет последовательность, включающую 2 тпн до и/или 1 тпн после известного гена сои в пределах 40 тпн от одного конца указанной негенной последовательности;
c) не содержит метилированный полинуклеотид;
d) не содержит 1 тпн последовательность, обладающую более чем 40% идентичностью последовательности с любой другой последовательностью в геноме сои; и
e) представляет подтверждение рекомбинации с частотой рекомбинации больше 0,01574 сМ/мпн.
В соответствии с одним вариантом осуществления предложена очищенная полинуклеотидная последовательность сои, включающая выбранную негенную последовательность. Выбранная негенная последовательность включает 2, 3, 4, 5, 6, 7 или 8 из следующих особенностей:
a) негенная последовательность не содержит метилированный полинуклеотид;
b) негенная последовательность демонстрирует частоту рекомбинации от 0,01574 до 83,52 сМ/мпн в геноме сои;
c) негенная последовательность демонстрирует уровень занятости нуклеосомами от 0 до 0,494 генома сои;
d) негенная последовательность обладает менее чем 40% идентичностью последовательности с любой другой 1 тпн последовательностью, содержащейся в геноме сои;
e) негенная последовательность имеет относительное значение местоположения от 0 до 0,99682 отношения геномного расстояния от центромеры хромосомы сои;
f) негенная последовательность имеет процентное содержание гуанина/цитозина в диапазоне от 14,36 до 45,9%;
g) негенная последовательность расположена проксимально к генной последовательности; и
h) 1 мпн область геномной последовательности сои, включающая указанную негенную последовательность, включает одну или более негенных последовательностей.
В соответствии с вариантом осуществления любые геномные последовательности сои, которые не представляют подтверждение рекомбинации с частотой рекомбинации больше 0,01574 сМ/мпн, исключаются из пула негенных геномных последовательностей сои.
В соответствии с одним вариантом осуществления выбранная негенная последовательность включает следующие особенности:
a) негенная последовательность не содержит больше 1% метилирования ДНК в последовательности
b) негенная последовательность имеет относительное значение местоположения от 0,211 до 0,976 отношения геномного расстояния от центромеры хромосомы сои;
c) негенная последовательность имеет процентное содержание гуанина/цитозина в диапазоне от 25,62 до 43,76%; и
d) негенная последовательность имеет длину от приблизительно 1 тпн до приблизительно 4,4 тпн.
КРАТКОЕ ОПИСАНИЕ ФИГУР
Фиг. 1. Представляет собой трехмерный график с 7018 отобранными геномными локусами, сгруппированными в 32 кластера. Кластеры могут быть изображены в виде трехмерного графика и обозначены цветом или другими индикаторами. Каждому кластеру был присвоен уникальный идентификатор для простоты визуализации, при этом все отобранные геномные локусы с одним и тем же идентификатором относились к одному кластеру. После процесса кластеризации репрезентативные отобранные геномные локусы отбирали из каждого кластера. Это выполняли, выбирая отобранные геномные локусы в каждом кластере, которые были наиболее близкими к центроиду этого кластера.
Фиг. 2. Представляет собой схематическое изображение, на котором показано хромосомное распределение оптимальных геномных локусов, выбранных как наиболее близких к центроиду каждого из 32 соответствующих кластеров.
Фиг. 3. Представляет собой схематическое изображение, на котором показано местоположение на хромосоме сои оптимальных геномных локусов, выбранных для подтверждения таргетинга.
Фиг. 4. Изображение универсальной донорной полинуклеотидной последовательности для интеграции посредством негомологичного соединения концов (NHEJ). Представлены два предполагаемых вектора, где целевая ДНК (ДНК X) включает один или более (то есть "1-N") сайтов связывания цинковых пальцев (ZFN BS) на обоих концах целевой ДНК. Вертикальные стрелки показывают уникальные сайты рестрикции, а горизонтальные стрелки представляют потенциальные участки ПЦР праймеров.
Фиг. 5. Изображение универсальной донорной полинуклеотидной последовательности для интеграции посредством направленной гомологией репарации (HDR). Целевая ДНК (ДНК X) включает две области гомологичных последовательностей (HA), фланкирующих целевую ДНК с сайтами связывания цинковых пальцев (ZFN), ограничивающими последовательности ДНК X и HA. Вертикальные стрелки показывают уникальные сайты рестрикции, а горизонтальные стрелки представляют потенциальные участки ПЦР праймеров.
Фиг. 6. Подтверждение выбранных геномных локусов-мишеней сои с использованием способа Быстрого анализа таргетинга (RTA) на основе NHEJ.
Фиг. 7. Карта плазмиды pDAB124280 (SEQ ID NO: 7561). Пронумерованные элементы (то есть GmPPL01ZF391R и GMPPL01ZF391L) соответствуют последовательностям связывания цинк-пальцевой нуклеазы длиной приблизительно 20-35 пар оснований, которые распознаются и расщепляются соответствующими белками, цинк-пальцевыми нуклеазами. Эти последовательности связывания цинковых пальцев и аннотируемая "последовательность UZI" (которая является 100-150 пн матричной областью, содержащей сайты рестрикции и последовательности ДНК для подбора праймеров или кодирующие последовательности) входят в универсальную донорную кассету. Также в эту схему плазмиды включены "104113 перекрывающиеся участки", которые являются последовательностями, которые обладают гомологией с плазмидным вектором для высокопроизводительной сборки универсальных донорных кассет в плазмидном векторе (то есть с помощью метода сборки Гибсона).
Фиг. 8. Карта плазмиды pDAB124281 (SEQ ID NO: 7562). Пронумерованные элементы (то есть GmPPL02ZF411R и GMPPL02ZF411L) соответствуют последовательностям связывания цинк-пальцевой нуклеазы длиной приблизительно 20-35 пар оснований, которые распознаются и расщепляются соответствующими белками, цинк-пальцевыми нуклеазами. Эти последовательности связывания цинковых пальцев и аннотируемая "последовательность UZI" (которая является 100-150 пн матричной областью, содержащей сайты рестрикции и последовательности ДНК для подбора праймеров или кодирующие последовательности) входят в универсальную донорную кассету. Также в эту схему плазмиды включены "104113 перекрывающиеся участки", которые являются последовательностями, которые обладают гомологией с плазмидным вектором для высокопроизводительной сборки универсальных донорных кассет в плазмидном векторе (то есть с помощью метода сборки Гибсона).
Фиг. 9. Карта плазмиды pDAB121278 (SEQ ID NO: 7563). Пронумерованные элементы (то есть GmPPL18_4 и GMPPL18_3) соответствуют последовательностям связывания цинк-пальцевой нуклеазы длиной приблизительно 20-35 пар оснований, которые распознаются и расщепляются соответствующими белками, цинк-пальцевыми нуклеазами. Эти последовательности связывания цинковых пальцев и аннотируемая "последовательность UZI" (которая является 100-150 пн матричной областью, содержащей сайты рестрикции и последовательности ДНК для подбора праймеров или кодирующие последовательности) входят в универсальную донорную кассету. Также в эту схему плазмиды включены "104113 перекрывающиеся участки", которые являются последовательностями, которые обладают гомологией с плазмидным вектором для высокопроизводительной сборки универсальных донорных кассет в плазмидном векторе (то есть с помощью метода сборки Гибсона).
Фиг. 10. Карта плазмиды pDAB123812 (SEQ ID NO: 7564). Пронумерованные элементы (то есть ZF538R и ZF538L) соответствуют последовательностям связывания цинк-пальцевой нуклеазы длиной приблизительно 20-35 пар оснований, которые распознаются и расщепляются соответствующими белками, цинк-пальцевыми нуклеазами. Эти последовательности связывания цинковых пальцев и аннотируемая "последовательность UZI" (которая является 100-150 пн матричной областью, содержащей сайты рестрикции и последовательности ДНК для подбора праймеров или кодирующие последовательности) входят в универсальную донорную кассету. Также в эту схему плазмиды включены "104113 перекрывающиеся участки", которые являются последовательностями, которые обладают гомологией с плазмидным вектором для высокопроизводительной сборки универсальных донорных кассет в плазмидном векторе (то есть с помощью метода сборки Гибсона).
Фиг. 11. Карта плазмиды pDAB121937 (SEQ ID NO: 7565). Пронумерованные элементы (то есть GmPPL34ZF598L, GmPPL34ZF598R, GmPPL36ZF599L, GmPPL36ZF599R, GmPPL36ZF600L и GmPPL36ZF600R) соответствуют последовательностям связывания цинк-пальцевой нуклеазы длиной приблизительно 20-35 пар оснований, которые распознаются и расщепляются соответствующими белками, цинк-пальцевыми нуклеазами. Эти последовательности связывания цинковых пальцев и аннотируемая "последовательность UZI" (которая является 100-150 пн матричной областью, содержащей сайты рестрикции и последовательности ДНК для подбора праймеров или кодирующие последовательности) входят в универсальную донорную кассету. Также в эту схему плазмиды включены "104113 перекрывающиеся участки", которые являются последовательностями, которые обладают гомологией с плазмидным вектором для высокопроизводительной сборки универсальных донорных кассет в плазмидном векторе (то есть с помощью метода сборки Гибсона).
Фиг. 12. Карта плазмиды pDAB123811 (SEQ ID NO: 7566). Пронумерованные элементы (то есть ZF 560L и ZF 560R) соответствуют последовательностям связывания цинк-пальцевой нуклеазы длиной приблизительно 20-35 пар оснований, которые распознаются и расщепляются соответствующими белками, цинк-пальцевыми нуклеазами. Эти последовательности связывания цинковых пальцев и аннотируемая "последовательность UZI" (которая является 100-150 пн матричной областью, содержащей сайты рестрикции и последовательности ДНК для подбора праймеров или кодирующие последовательности) входят в универсальную донорную кассету. Также в эту схему плазмиды включены "104113 перекрывающиеся участки", которые являются последовательностями, которые обладают гомологией с плазмидным вектором для высокопроизводительной сборки универсальных донорных кассет в плазмидном векторе (то есть с помощью метода сборки Гибсона).
Фиг. 13. Карта плазмиды pDAB124864 (SEQ ID NO: 7567). Пронумерованные элементы (то есть ZF631L и ZF631R) соответствуют последовательностям связывания цинк-пальцевой нуклеазы длиной приблизительно 20-35 пар оснований, которые распознаются и расщепляются соответствующими белками, цинк-пальцевыми нуклеазами. Эти последовательности связывания цинковых пальцев и аннотируемая "последовательность UZI" (которая является 100-150 пн матричной областью, содержащей сайты рестрикции и последовательности ДНК для подбора праймеров или кодирующие последовательности) входят в универсальную донорную кассету. Также в эту схему плазмиды включены "104113 перекрывающиеся участки", которые являются последовательностями, которые обладают гомологией с плазмидным вектором для высокопроизводительной сборки универсальных донорных кассет в плазмидном векторе (то есть с помощью метода сборки Гибсона).
Фиг. 14. Карта плазмиды pDAB7221 (SEQ ID NO: 7569). Эта плазмида содержит промотор вируса мозаики жилок маниока (CsVMV), направляющий экспрессию белка GFP и фланкированный Agrobacterium tumefaciens (AtuORF 24 3’UTR).
Фиг. 15A-15C. Гистограмма показателей (длина, экспрессия кодирующей области в пределах 40 тпн локусов и частота рекомбинации) для идентифицированных оптимальных негенных локусов сои. На Фиг. 15A показано распределение длин полинуклеотидных последовательностей оптимальных геномных локусов (OGL). На Фиг. 15B показано распределение оптимальных негенных локусов кукурузы относительно их частоты рекомбинации. На Фиг. 15C показано распределение экспрессируемых последовательностей нуклеиновых кислот по их близости (логарифмическая шкала) к оптимальным геномным локусам (OGL).
ПОДРОБНОЕ ОПИСАНИЕ
ОПРЕДЕЛЕНИЯ
В описании и формуле изобретения будет использоваться следующая терминология в соответствии с определениями, представленными ниже.
Термин "приблизительно", при использовании в настоящем описании, означает больше или меньше чем указанное значение или диапазон значений на 10 процентов, но при этом не предполагается, что любое значение или диапазон значений определяются только этим более широким определением. Каждое значение или диапазон значений, перед которыми указан термин "приблизительно", также должны охватывать вариант указанного абсолютного значения или диапазона значений.
При использовании в настоящем описании термин "растение" включает целое растение и любое потомство, клетку, ткань или часть растения. Термин "части растения" включает любую часть(и) растения, в том числе, например, и без ограничения: семя (включая зрелое семя и незрелое семя); черенки растения; клетку растения; культуру клеток растения; орган растения (например, пыльцу, зародыши, цветки, плоды, побеги, листья, корни, стебли и экспланты). Ткань растения или орган растения могут быть семенем, каллусом или любой другой группой клеток растения, которые организованы в структурную или функциональную единицу. Растительная клетка или культура тканей могут быть способны к регенерации растения, обладающего физиологическими и морфологическими признаками растения, из которого была получена клетка или ткань, и регенерации растения, имеющего по существу такой же генотип, что и указанное растение. Напротив, некоторые растительные клетки не способны к регенерации с получением растений. Регенерируемые клетки в растительной клетке или культуре тканей могут быть зародышами, протопластами, меристематическими клетками, каллусом, пыльцой, листьями, пыльниками, корнями, корневыми кончиками, пестиками початков, цветками, зернами, початками, стержнями початков, обверткой початков или стеблями.
Части растения включают пригодные для сбора части и части, пригодные для размножения потомства растений. Части растения, пригодные для размножения, включают, например, и без ограничения: семя; плод; черенок; сеянец; клубень и корневище. Пригодная для сбора часть растения может быть любой полезной частью растения, включая, например, и без ограничения: цветок; пыльцу; сеянец; клубень; лист; стебель; плод; семя и корень.
Растительная клетка является структурной и физиологической единицей растения. Растительные клетки, при использовании в настоящем описании, включают протопласты и протопласты с клеточной стенкой. Растительная клетка может быть в форме выделенной одиночной клетки или агрегата клеток (например, рыхлого каллуса и культивируемой клетки) и может быть частью более высокоорганизованной единицы (например, растительной ткани, органа растения и растения). Таким образом, растительная клетка может быть протопластом, гаметообразующей клеткой или клеткой или коллекцией клеток, которые могут регенерировать с образованием целого растения. Таким образом, семя, которое включает множество растительных клеток и способно к регенерации с образованием целого растения, считается "частью растения" в вариантах осуществления в настоящей заявке.
Термин "протопласт", при использовании в настоящем описании, относится к растительной клетке, у которой была полностью или частично удалена ее клеточная стенка, и ее липидная бислойная мембрана была обнажена. Как правило, протопласт представляет собой выделенную растительную клетку без клеточных стенок, которая обладает возможностью регенерации с получением клеточной культуры или целого растения.
При использовании в настоящем описании термины "нативный" или "природный" определяют состояние, существующее в природе. "Нативная последовательность ДНК" является последовательностью ДНК, присутствующей в природе, которая была получена естественными способами или с помощью традиционных методик селекции, но не была получена с помощью генной инженерии (например, с использованием методик молекулярной биологии/ трансформации).
При использовании в настоящем описании, "эндогенная последовательность" определяет нативную форму полинуклеотида, гена или полипептида в его естественном местоположении в организме или в геноме организма.
Термин "выделенный", при использовании в настоящем описании, означает удаленный из своего естественного окружения.
Термин "очищенный", при использовании в настоящем описании, относится к выделению молекулы или соединения в форме, которая по существу не содержит контаминирующих примесей, обычно связанных с молекулой или соединением в нативном или естественном окружении, и означает повышение чистоты в результате отделения от других компонентов исходной композиции. Термин "очищенная нуклеиновая кислота" используется в настоящем описании для описания последовательности нуклеиновой кислоты, которая была отделена от других соединений, в том числе, без ограничения перечисленными, полипептидов, липидов и углеводов.
Термины "полипептид", "пептид" и "белок" используются попеременно для обозначения полимера из аминокислотных остатков. Термин также относится к полимерам аминокислот, в которых одна или больше аминокислот являются химическими аналогами или модифицированными производными соответствующих природных аминокислот.
При использовании в настоящем описании "оптимальные геномные локусы двудольного растения", "оптимальные негенные локусы двудольного растения", "оптимальные негенные локусы" или "оптимальные геномные локусы (OGL)" являются нативной последовательностью ДНК, присутствующей в ядерном геноме двудольного растения, которая обладает следующими свойствами: негенная, гипометилированная, может служить мишенью и находится в проксимальном положении относительно генной области, где геномная область вокруг оптимальных геномных локусов двудольного растения представляет подтверждение рекомбинации.
При использовании в настоящем описании термины "оптимальные геномные локусы сои", "оптимальные негенные локусы сои", "оптимальные негенные локусы" или "оптимальные геномные локусы (OGL)" используются попеременно для обозначения нативной последовательности ДНК, присутствующей в ядерном геноме двудольного растения, которая обладает следующими свойствами: негенная, гипометилированная, может служить мишенью и находится в проксимальном положении относительно генной области, где геномная область вокруг оптимальных геномных локусов двудольного растения представляет подтверждение рекомбинации.
При использовании в настоящем описании термины "негенная последовательность двудольного растения" или "негенная геномная последовательность двудольного растения" используются попеременно для обозначения нативной последовательности ДНК, присутствующей в ядерном геноме двудольного растения, имеющей длину по меньшей мере 1 тпн и не содержащей никаких открытых рамок считывания, генных последовательностей или регуляторных последовательностей генов. Кроме того, негенная последовательность двудольного растения не включает последовательность интрона (то есть интроны исключены из определения негенный). Негенная последовательность не может транскрибироваться или транслироваться в белок. Геномы многих растений содержат негенные области. До 95% генома могут быть негенным, причем такие области могут состоять главным образом из ДНК с повторяющимися последовательностями.
При использовании в настоящем описании термины "негенная последовательность сои" или "негенная геномная последовательность сои" используются попеременно для обозначения нативной последовательности ДНК, присутствующей в ядерном геноме растения сои, имеющей длину по меньшей мере 1 тпн и не содержащей никаких открытых рамок считывания, генных последовательностей или регуляторных последовательностей генов. Кроме того, негенная последовательность сои не включает последовательность интрона (то есть интроны исключены из определения негенный). Негенная последовательность не может транскрибироваться или транслироваться в белок. Геномы многих растений содержат негенные области. До 95% генома может быть негенным, причем эти области могут состоять главным образом из ДНК с повторяющимися последовательностями.
При использовании в настоящем описании, "генная область" определяется как полинуклеотидная последовательность, которая включает открытую рамку считывания, кодирующую РНК и/или полипептид. Генная область также может охватывать любые идентифицируемые смежные 5′ и 3′ некодирующие нуклеотидные последовательности, участвующие в регуляции экспрессии открытой рамки считывания, расположенные на протяжении до приблизительно 2 тпн перед кодирующей областью и 1 тпн после кодирующей области, но, возможно, дальше перед ней или после нее. Генная область также включает любые интроны, которые могут присутствовать в генной области. Кроме того, генная область может включать одну генную последовательность или множество генных последовательностей, которые чередуются с короткими промежутками (меньше 1 тпн) негенных последовательностей.
При использовании в настоящем описании "целевая нуклеиновая кислота", "целевая ДНК" или "донор" определяются как последовательность нуклеиновой кислоты/ДНК, которая была выбрана для сайт-специфической, направленной вставки в геном двудольного растения, например геном сои. Целевая нуклеиновая кислота может иметь любую длину, например длину от 2 до 50000 нуклеотидов (или любое целочисленное значение в этом или указанном выше диапазоне), предпочтительно длину приблизительно от 1000 до 5000 нуклеотидов (или любое целочисленное значение в этом диапазоне). Целевая нуклеиновая кислота может включать одну или более кассет экспрессии гена, которые дополнительно включают активно транскрибируемые и/или транслируемые генные последовательности. С другой стороны, целевая нуклеиновая кислота может включать полинуклеотидную последовательность, которая не включает функциональную кассету экспрессии гена или весь ген (например, может просто включать регуляторные последовательности, такие как промотор) или, возможно, не содержит идентифицируемых элементов экспрессии гена или какую-либо активно транскрибируемую генную последовательность. Целевая нуклеиновая кислота необязательно может содержать аналитический домен. При вставке целевой нуклеиновой кислоты в геном двудольного растения, например сои, вставленные последовательности именуются как "вставленная целевая ДНК". Кроме того, целевая нуклеиновая кислота может быть ДНК или РНК, может быть линейной или кольцевой и может быть одноцепочечной или двухцепочечной. Она может быть введена в клетку в виде голой нуклеиновой кислоты, в виде комплекса с одним или более средствами доставки (например, липосомами, полоксамерами, T-цепью, инкапсулированной с белками, и т.д.) или содержаться в бактериальном или вирусном носителе, таком как, например, Agrobacterium tumefaciens или аденовирус или адено-ассоциированный вирус (AAV), соответственно.
При использовании в настоящем описании термин "аналитический домен" определяет последовательность нуклеиновой кислоты, которая содержит функциональные элементы, которые способствуют направленной вставке последовательностей нуклеиновых кислот. Например, аналитический домен может содержать специальные сайты рестрикции, сайты связывания цинковых пальцев, сконструированные посадочные участки или сконструированные платформы интеграции трансгена, и может включать или не включать регуляторные элементы гена или открытую рамку считывания. См., например, патентную публикацию США 20110191899, полностью включенную в настоящую заявку посредством отсылки.
При использовании в настоящем описании термин "выбранная последовательность двудольного растения" определяет нативную геномную последовательность ДНК двудольного растения, которая была выбрана для анализа с целью определения, подходит ли данная последовательность в качестве оптимальных негенных геномных локусов двудольного растения.
При использовании в настоящем описании термин "выбранная последовательность сои" определяет нативную геномную последовательность ДНК растения сои, которая была выбрана для анализа с целью определения, подходит ли данная последовательность в качестве оптимальных негенных геномных локусов сои.
При использовании в настоящем описании термин "гипометилирование" или "гипометилированный", в отношении последовательности ДНК, определяет состояние пониженного метилирования нуклеотидных остатков ДНК в данной последовательности ДНК. Как правило, пониженное метилирование относится к количеству метилированных остатков аденина или цитозина по отношению к среднему уровню метилирования в негенных последовательностях, содержащихся в геноме двудольного растения, такого как растение сои.
При использовании в настоящем описании "последовательность-мишень" является полинуклеотидной последовательностью, которая достаточно уникальна в ядерном геноме, чтобы обеспечивать возможность сайт-специфической, направленной вставки целевой нуклеиновой кислоты в одну определенную последовательность.
При использовании в настоящем описании термин "неповторяющаяся" последовательность определяется как последовательность длиной по меньшей мере 1 тпн, которая обладает менее чем 40% идентичностью с любой другой последовательностью в геноме двудольного растения, такого как соя. Вычисления идентичности последовательности могут быть определены при использовании любой стандартной методики, известной специалистам в данной области техники, включающей, например, сканирование выбранной геномной последовательности в геноме двудольного растения, например, в геноме сои сорта Williams82, при использовании поиска гомологии на основе BLASTTM, с использованием программы NCBI BLASTTM+ (версия 2.2.25), запущенной с использованием параметров настройки по умолчанию (Stephen F. Altschul et al (1997), "Gapped BLAST and PSI-BLAST: a new generation of protein database search programs", Nucleic Acids Res. 25:3389-3402). Например, в результате анализа выбранных последовательностей сои (из генома Glycine max сорта Williams82) первое совпадение в BLASTTM, идентифицированное при таком поиске, представляет последовательность двудольного растения, например, последовательность сои сорта Williams82, непосредственно. Второе совпадение в BLASTTM идентифицировали для каждой выбранной последовательности сои и охват выравнивания (представленный как процент выбранной последовательности сои, занятой совпадающей последовательностью из BLASTTM) совпадающей последовательности использовали в качестве показателя уникальности выбранной последовательности сои в геноме двудольного растения, такого как соя. Эти значения охвата выравнивания для второго совпадения в BLASTTM варьировали от минимума 0% до максимума 39,97% идентичности последовательности. Любые последовательности, которые выравнивали с более высокими уровнями идентичности последовательности, не рассматривали.
Термин "в проксимальном положении к генной области", в случае использования в отношении негенной последовательности, определяет относительное местоположение негенной последовательности к генной области. В частности, анализируют количество генных областей в пределах соседней 40 тпн области (то есть в пределах 40 тпн на любом конце выбранной последовательности оптимальных геномных локусов сои). Этот анализ был завершен исследованием данных аннотации генов и местоположения известных генов в геноме известного двудольного растения, такого как соя, которые были получены из базы геномов однодольных растений, например, из Базы данных генома сои. Для каждого из оптимальных негенных геномных локусов сои, например, 7018 оптимальных негенных геномных локусов сои, было определено 40 тпн окно вокруг последовательности оптимальных геномных локусов, после чего подсчитали количество аннотируемых генов, местоположения которых накладывались на это окно. Количество генных областей варьировало от минимум 1 гена до максимум 18 генов в пределах соседней 40 тпн области.
Термин "известная кодирующая последовательность сои", при использовании в настоящем описании, относится к любой полинуклеотидной последовательности, идентифицированной из любой геномной базы данных двудольных растений, включая Геномную базу данных сои (Soybean Genomic Database (www.soybase.org, Shoemaker, R.C. et al. SoyBase, the USDA-ARS soybean genetics and genomics database. Nucleic Acids Res. 2010 Jan; 38 (Database issue):D843-6), которая включает открытую рамку считывания, до или после процессинга последовательностей интронов, и транскрибируется в мРНК и необязательно транслируется в последовательность белка при помещении под контроль подходящих генетических регуляторных элементов. Известной кодирующей последовательностью сои может быть последовательность кДНК или геномная последовательность. В некоторых случаях известная кодирующая последовательность сои может быть аннотирована как функциональный белок. В других случаях известная кодирующая последовательность сои может быть не аннотирована.
Термин "предсказанная кодирующая последовательность двудольного растения", при использовании в настоящем описании, относится к любым экспрессируемым маркерным полинуклеотидным последовательностям (EST), описанным в геномной базе данных двудольных растений, например, в геномной базе данных сои. Последовательности EST идентифицируют из библиотек кДНК, конструируемых с использованием олиго(дТ) праймеров, для направления синтеза первой цепи с использованием обратной транскриптазы. Получаемые в результате последовательности EST представляют собой считываемые фрагменты однонаправленного секвенирования длиной меньше 500 пн, полученные с 5′ или 3′ конца вставки кДНК. Множественные EST могут быть выровнены с получением одного контига. Идентифицированные последовательности EST загружают в геномную базу данных двудольных растений, например геномную базу данных сои, после чего можно проводить поиск с помощью биоинформационных методов для предсказания соответствующих геномных полинуклеотидных последовательностей, которые включают кодирующую последовательность, которая транскрибируется в мРНК и необязательно транслируется в последовательность белка, если она находится под контролем подходящих генетических регуляторных элементов.
Термин "предсказанная кодирующая последовательность сои", при использовании в настоящем описании, относится к любым экспрессируемым маркерным полинуклеотидным последовательностям (EST), описанным в геномной базе данных сои, например Геномной базе данных сои. Последовательности EST идентифицируют из библиотек кДНК, полученных с использованием олиго(дТ) праймеров для направления синтеза первой цепи обратной транскриптазой. Получаемые в результате последовательности EST представляют собой считываемые фрагменты однонаправленного секвенирования длиной меньше 500 пн, полученные с 5′ или 3′ конца вставки кДНК. Множественные EST могут быть выровнены с получением одного контига. Идентифицированные последовательности EST загружают в геномную базу данных сои, например Геномную базу данных сои, после чего можно проводить поиск с помощью биоинформационных методов для предсказания соответствующих геномных полинуклеотидных последовательностей, которые включают кодирующую последовательность, которая транскрибируется в мРНК и необязательно транслируется в последовательность белка, если она находится под контролем подходящих генетических регуляторных элементов.
Термин "подтверждение рекомбинации", при использовании в настоящем описании, относится к частотам мейотической рекомбинации между любой парой геномных маркеров двудольного растения, например геномных маркеров сои, через область хромосомы, включающую выбранную последовательность сои. Частоты рекомбинации вычисляли на основе отношения генетического расстояния между маркерами (в сантиморганах (сМ)) к физическому расстоянию между маркерами (в миллионах пар нуклеотидов (мпн)). Чтобы выбранная последовательность сои имела подтверждение рекомбинации, выбранная последовательность сои должна содержать по меньшей мере одно событие рекомбинации между двумя маркерами, фланкирующими выбранную последовательность сои, как обнаруживают с использованием набора данных высокого разрешения для маркеров, полученного из популяций множественного картирования.
При использовании в настоящем описании термин "относительное значение местоположения" является вычисленным значением, определяющим расстояние геномного локуса от его соответствующей центромеры хромосомы. Для каждой выбранной последовательности сои измеряют геномное расстояние (в пн) от нативного местоположения выбранной последовательности сои до центромеры хромосомы, на которой она расположена. Относительное местоположение выбранной последовательности сои на хромосоме представляют как отношение ее геномного расстояния до центромеры к длине определенного хромосомного плеча (измеряемой в пн), на котором она находится. Эти относительные значения местоположения для оптимальных негенных геномных локусов сои могут быть получены для различных двудольных растений, относительные значения местоположения для набора данных сои варьируют от минимум 0 до максимум 0,99682 отношения геномного расстояния.
Термин "экзогенная последовательность ДНК", при использовании в настоящем описании, является любой последовательностью нуклеиновой кислоты, которая была удалена из ее нативного местоположения и вставлена в новое местоположение, с изменением последовательностей, которые фланкируют указанную последовательность нуклеиновой кислоты, которая была перемещена. Например, экзогенная последовательность ДНК может включать последовательность из другого биологического вида.
"Связывание" относится к сиквенс-специфическому взаимодействию между макромолекулами (например, между белком и нуклеиновой кислотой). Не все компоненты связывающего взаимодействия должны быть сиквенс-специфическими (например, контакты с фосфатными остатками в основной цепи ДНК), при условии, что взаимодействие в целом является сиквенс-специфическим. Такие взаимодействия обычно характеризуются константой диссоциации (Kd). "Аффинность" относится к силе связывания: увеличение аффинности связывания соответствует более низкой константе связывания (Kd).
"Связывающий белок" является белком, который способен связываться с другой молекулой. Связывающий белок может связываться, например, с молекулой ДНК (ДНК-связывающий белок), молекулой РНК (РНК-связывающий белок) и/или молекулой белка (белок-связывающий белок). В случае белок-связывающего белка он может связываться сам с собой (с образованием гомодимеров, гомотримеров и т.д.), и/или он может связываться с одной или более молекулами другого белка или белков. Связывающий белок может иметь больше одного типа связывающей активности. Например, цинк-пальцевые белки обладают ДНК-связывающей, РНК-связывающей и белок-связывающей активностью.
При использовании в настоящем описании термин "цинковые пальцы" определяет области аминокислотной последовательности в связывающем домене ДНК-связывающего белка, структура которого стабилизируется при координационном взаимодействии с ионом цинка.
"ДНК-связывающий белок с цинковыми пальцами" (или связывающий домен) является белком или доменом более крупного белка, который сиквенс-специфически связывает ДНК посредством одного или нескольких цинковых пальцев, которые являются областями аминокислотной последовательности в связывающем домене, структура которых стабилизируется при координационном взаимодействии с ионом цинка. Термин ДНК-связывающий белок с цинковыми пальцами часто сокращенно называют белком с цинковыми пальцами или ZFP. Связывающие домены с цинковыми пальцами могут быть "сконструированы" для связывания с заданной нуклеотидной последовательностью. Неограничивающими примерами способов создания белков с цинковыми пальцами являются конструирование и отбор. Сконструированный белок с цинковыми пальцами представляет собой белок, не встречающийся в природе, конструкция/состав которого преимущественно является результатом рациональных критериев. Рациональные критерии конструирования включают применение правил замен и компьютерных алгоритмов для обработки информации из базы данных, в которой хранится информация о существующих конструкциях ZFP и их данные связывания. См., например, патенты США 6,140,081; 6,453,242; 6,534,261 и 6,794,136; см. также WO 98/53058; WO 98/53059; WO 98/53060; WO 02/016536 и WO 03/016496.
"ДНК-связывающий домен TALE" или "TALE" является полипептидом, включающим один или более доменов/повторяющихся звеньев TALE. Повторяющиеся домены участвуют в связывании TALE с его когнатной целевой последовательностью ДНК. Одиночное "повторяющееся звено" (также называемое "повтором") обычно имеет длину 33-35 аминокислот и демонстрирует, по меньшей мере, некоторую гомологию последовательности с другими последовательностями повторов TALE в природном белке TALE. См., например, патентную публикацию США 20110301073, полностью включенную в настоящую заявку посредством отсылки.
CRISPR (от англ. clustered regularly interspaced short palindromic repeats - кластерные, разделенные регулярными интервалами, короткие палиндромные повторы)/Cas (CRISPR-ассоциированная нуклеазная система. Коротко, "ДНК-связывающий домен CRISPR" является молекулой РНК с короткой цепью, которая, действуя в сочетании с ферментом Cas, может селективно распознавать, связывать и расщеплять геномную ДНК. Система CRISPR/Cas может быть сконструирована для создания двухцепочечных разрывов (DSB) в требуемой мишени в геноме, при этом на репарацию DSB может влиять использование ингибиторов репарации для усиления репарации пониженной точности. См., например, Jinek et al (2012) Science 337, стр. 816-821, Jinek et al, (2013), eLife 2:e00471, и David Segal, (2013) eLife 2:e00563).
Цинк-пальцевые, CRISPR и TALE связывающие домены могут быть "сконструированы" для связывания с заданной нуклеотидной последовательностью, например, посредством инженерии (изменения одной или более аминокислот) в области спирали распознавания природного цинкового пальца. Аналогичным образом, TALE могут быть "сконструированы" для связывания с заданной нуклеотидной последовательностью, например, посредством инженерии аминокислот, участвующих в связывании ДНК (повторяющаяся область с двумя вариабельными остатками или область RVD (от англ. - repeat variable diresidue)). Таким образом, сконструированные ДНК-связывающие белки (цинк-пальцевые или TALE) являются белками, которые не встречаются в природе. Неограничивающими примерами способов инженерии ДНК-связывающих белков являются конструирование и отбор. Сконструированный ДНК-связывающий белок представляет собой белок, не встречающийся в природе, конструкция/состав которого преимущественно является результатом рациональных критериев. Рациональные критерии конструирования включают применение правил замены и компьютерных алгоритмов для обработки информации из базы данных, в которой хранится информация о существующих конструкциях ZFP и/или TALE и их данные связывания. См., например, патенты США 6,140,081; 6,453,242 и 6,534,261; см. также WO 98/53058; WO 98/53059; WO 98/53060; WO 02/016536 и WO 03/016496, и публикации США 20110301073, 20110239315 и 20119145940.
"Выбранный" белок с цинковыми пальцами, CRISPR или TALE представляет собой белок, не существующий в природе, получение которого, прежде всего, является результатом эмпирического процесса, такого как фаговый дисплей, ловушка взаимодействий или отбор гибридов. См. например, патенты США 5,789,538; US 5,925,523; US 6,007,988; US 6,013,453; US 6,200,759; WO 95/19431; WO 96/06166; WO 98/53057; WO 98/54311; WO 00/27878; WO 01/60970 WO 01/88197 и WO 02/099084, и публикации США 20110301073, 20110239315 и 20119145940.
"Рекомбинация" относится к процессу обмена генетической информацией между двумя полинуклеотидами, включающему, без ограничения перечисленным, донорный захват при негомологичном соединении концов (NHEJ) и гомологичную рекомбинацию. В рамках настоящего описания, "гомологичная рекомбинация (ГР)" относится к специализированной форме такого обмена, который происходит, например, во время репарации двухцепочечных разрывов в клетках, осуществляемой посредством направленных гомологией механизмов репарации. Этот процесс требует гомологии нуклеотидной последовательности, использует "донорную" молекулу для матричной репарации молекулы "мишени" (то есть молекулы, в которой образовался двухцепочечный разрыв) и известен под разными названиями, такими как "некроссоверная конверсия генов" или "конверсия генов на коротких участках", поскольку он приводит к переносу генетической информации от донора к мишени. Без желания быть связанными какой-либо конкретной теорией, предполагают, что такой перенос может включать коррекцию неспаренных оснований гетеродуплекса ДНК, который образуется между расщепленной мишенью и донором, и/или "синтез-зависимый отжиг цепей", в котором донор используется для восстановления генетической информации, которая становится частью мишени, и/или подобные процессы. Такая специализированная ГР часто приводит к изменению последовательности молекулы-мишени, в результате чего часть или вся последовательность донорного полинуклеотида встраивается в целевой полинуклеотид. В отношении ГР-направленной интеграции, донорная молекула содержит по меньшей мере 2 области гомологии с геномом ("плечи гомологии") длиной по меньшей мере 50-100 пар оснований. См., например, патентную публикацию США 20110281361.
В способах настоящего описания одна или более направленных нуклеаз, как описано в настоящей заявке, создают двухцепочечный разрыв в целевой последовательности (например, клеточного хроматина) на заданном участке, при этом "донорный" полинуклеотид, обладающий гомологией с нуклеотидной последовательностью в области разрыва для ГР-опосредованной интеграции или не обладающий гомологией с нуклеотидной последовательностью в области разрыва для NHEJ-опосредованной интеграции, может быть введен в клетку. Присутствие двухцепочечного разрыва, как было показано, способствует интеграции донорной последовательности. Донорная последовательность может быть интегрирована физически или, в альтернативе, донорный полинуклеотид используется в качестве матрицы для репарации разрыва посредством гомологичной рекомбинации, что приводит к введению полной или части нуклеотидной последовательности, как в доноре, в клеточный хроматин. Таким образом, первая последовательность в клеточном хроматине может быть изменена и, в некоторых вариантах осуществления, может быть превращена в последовательность, присутствующую в донорном полинуклеотиде. Таким образом, использование терминов "заменяет" или "замена", как можно понимать, представляет замену одной нуклеотидной последовательности другой (то есть замену последовательности в информационном смысле) и не требует обязательно физической или химической замены одного полинуклеотида другим.
В любом из способов, описанных в настоящей заявке, дополнительные пары белков с цинковыми пальцами, CRISPRS или TALEN могут использоваться для дополнительного двухцепочечного расщепления дополнительных целевых участков в клетке.
Любой из способов, описанных в настоящей заявке, может применяться для вставки донора любого размера и/или частичной или полной инактивации одной или более последовательностей-мишеней в клетке посредством направленной интеграции донорной последовательности, которая прерывает экспрессию целевого гена(ов). Также предложены клеточные линии с частично или полностью инактивированными генами.
Кроме того, способы направленной интеграции, описанные в настоящей заявке, также могут применяться для интеграции одной или более экзогенных последовательностей. Экзогенная последовательность нуклеиновой кислоты может включать, например, один или более генов или молекул кДНК, или любой тип кодирующей или некодирующей последовательности, а также один или более элементов контроля (например, промоторы). Кроме того, экзогенная последовательность нуклеиновой кислоты (трансген) может давать одну или более молекул РНК (например, малые шпилечные РНК (мшРНК), ингибиторные РНК (РНКи), микроРНК (миРНК) и т.д.) или белок.
"Расщепление", при использовании в настоящем описании, определяет расщепление фосфатно-сахарной основной цепи молекулы ДНК. Расщепление может быть инициировано множеством методов, включающих, без ограничения перечисленными, ферментативный или химический гидролиз фосфодиэфирной связи. Возможно как одноцепочечное расщепление, так и двухцепочечное расщепление, причем двухцепочечное расщепление может происходить в результате двух отдельных событий одноцепочечного расщепления. Расщепление ДНК может приводить к образованию тупых концов или ступенчатых концов. В некоторых вариантах осуществления слитые полипептиды используются для направленного двухцепочечного расщепления ДНК. "Расщепляющий домен" включает одну или более полипептидных последовательностей, которые обладают каталитической активностью для расщепления ДНК. Расщепляющий домен может содержаться в одиночной полипептидной цепи, или расщепляющая активность может являться результатом ассоциации двух (или более) полипептидов.
"Расщепляющий полудомен" является полипептидной последовательностью, которая в сочетании со вторым полипептидом (идентичным или другим) образует комплекс, обладающий расщепляющей активностью (предпочтительно расщепляющей активностью в отношении двойной цепи). Термины "первый и второй расщепляющие полудомены", "+ и – расщепляющие полудомены" и "правый и левый расщепляющие полудомены" используются попеременно для обозначения пар расщепляющих полудоменов, которые димеризуются.
"Сконструированный расщепляющий полудомен" является расщепляющим полудоменом, который был модифицирован с получением облигатных гетеродимеров с другим расщепляющим полудоменом (например, другим сконструированным расщепляющим полудоменом). См., также, патентные публикации США 2005/0064474, 20070218528, 2008/0131962 и 2011/0201055, полностью включенные в настоящее описание посредством отсылки.
"Сайт-мишень" или "последовательность-мишень" относится к части нуклеиновой кислоты, с которой связывается связывающая молекула, при условии, что существуют достаточные условия для связывания.
Нуклеиновые кислоты включают ДНК и РНК, могут быть одно- или двухцепочечными; могут быть линейными, разветвленными или кольцевыми; и могут иметь любую длину. Нуклеиновые кислоты включают нуклеиновые кислоты, способные к образованию дуплексов, а также триплекс-образующие нуклеиновые кислоты. См., например, патенты США 5,176,996 и 5,422,251. Белки включают, без ограничения перечисленными, ДНК-связывающие белки, факторы транскрипции, факторы ремоделирования хроматина, связывающие метилированную ДНК белки, полимеразы, метилазы, деметилазы, ацетилазы, деацетилазы, киназы, фосфатазы, интегразы, рекомбиназы, лигазы, топоизомеразы, гиразы и геликазы.
"Продукт экзогенной нуклеиновой кислоты" включает полинуклеотидные и полипептидные продукты, например, продукты транскрипции (полинуклеотиды, такие как РНК) и продукты трансляции (полипептиды).
"Слитая" молекула является молекулой, в которой связаны две или более молекул-субъединиц, например, ковалентно. Молекулы-субъединицы могут быть молекулами одного химического типа или могут быть молекулами разных химических типов. Примеры первого типа слитой молекулы включают, без ограничения перечисленными, слитые белки (например, слитые ZFP ДНК-связывающий домен и расщепляющий домен) и слитые нуклеиновые кислоты (например, нуклеиновая кислота, кодирующая слитый белок, описанный выше). Примеры второго типа слитой молекулы включают, без ограничения перечисленными, слитые триплекс-образующую нуклеиновую кислоту и полипептид, и слитые белок, связывающий малую борозду, и нуклеиновую кислоту. Экспрессия слитого белка в клетке может являться результатом доставки слитого белка в клетку или доставки полинуклеотида, кодирующего слитый белок, в клетку, где полинуклеотид транскрибируется и транслируется в транскрипт с получением слитого белка. Транс-сплайсинг, расщепление полипептида и лигирование полипептида также могут быть включены в экспрессию белка в клетке. Методы доставки полинуклеотида и полипептида в клетки представлены в другой части настоящего описания.
В рамках настоящего описания, "ген" включает область ДНК, кодирующую продукт гена (см. ниже), а также все области ДНК, которые регулируют синтез продукта гена, независимо от того, примыкают ли такие регуляторные последовательности к кодирующим и/или транскрибируемым последовательностям, или функционально связаны с ними, или нет. Таким образом, ген включает промоторные последовательности, терминаторы, последовательности регуляции трансляции, такие как участки связывания рибосом и участки внутренней посадки рибосомы, энхансеры, сайленсеры, инсуляторы, граничные элементы, точки начала репликации, участки связывания с матриксом и области контроля локусов, но не должен ограничиваться ими.
"Экспрессия гена" относится к преобразованию информации, содержащейся в гене, в продукт гена. Продукт гена может представлять собой продукт непосредственной транскрипции гена (например, мРНК, тРНК, рРНК, антисмысловая РНК, интерферирующая РНК, рибозим, структурная РНК или РНК любого другого типа) или белок, синтезируемый при трансляции мРНК. Продукты генов также включают РНК, модифицируемые в результате таких процессов, как кэпирование, полиаденилирование, метилирование и редактирование, и белки, модифицируемые, например, в результате метилирования, ацетилирования, фосфорилирования, убиквитинирования, АДФ-рибозилирования, миристилирования и гликозилирования.
Идентичность последовательности: термин "идентичность последовательности" или "идентичность", при использовании в настоящем описании в отношении двух полинуклеотидных или полипептидных последовательностей, относится к остаткам в двух последовательностях, которые являются одинаковыми при выравнивании с максимальным соответствием в указанном окне сравнения.
При использовании в настоящем описании, термин "процент идентичности последовательности" относится к значению, определяемому при сравнении двух оптимально выровненных последовательностей (например, последовательностей нуклеиновых кислот и аминокислотных последовательностей) в окне сравнения, где часть последовательности в окне сравнения может включать добавления или делеции (то есть пропуски) по сравнению с референсной последовательностью (которая не включает добавления или делеции) для оптимального выравнивания двух указанных последовательностей. Процент вычисляют путем определения количества положений, в которых идентичный нуклеотидный или аминокислотный остаток встречается в обеих последовательностях, с получением количества совпадающих положений, деления количества совпадающих положений на общее количество положений в окне сравнения, и умножения результата на 100, с получением процента идентичности последовательности.
Методы выравнивания последовательностей для сравнения известны в уровне техники. Различные программы и алгоритмы выравнивания описаны, например, в: Smith and Waterman (1981) Adv. Appl. Math. 2:482; Needleman and Wunsch (1970) J. Mol. Biol. 48:443; Pearson and Lipman (1988) Proc. Natl. Acad. Sci. U.S.A. 85:2444; Higgins and Sharp (1988) Gene 73:237-44; Higgins and Sharp (1989) CABIOS 5:151-3; Corpet et al. (1988) Nucleic Acids Res. 16:10881-90; Huang et al. (1992) Comp. Appl. Biosci. 8:155-65; Pearson et al. (1994) Methods Mol. Biol. 24:307-31; Tatiana et al. (1999) FEMS Microbiol. Lett. 174:247-50. Подробное рассмотрение методов выравнивания последовательностей и вычисления гомологии можно найти, например, в Altschul et al. (1990) J. Mol. Biol. 215:403-10. Средство поиска основного локального выравнивания (BLASTTM; Altschul et al. (1990)) Национального центра биотехнологической информации (NCBI) доступно из нескольких источников, включая Национальный центр биотехнологической информации (Bethesda, MD), и в Интернете, для использования в сочетании с несколькими программами для анализа последовательностей. Описание того, как определить идентичность последовательности с использованием этой программы, доступно в Интернете в разделе "help" BLASTTM. Для сравнений последовательностей нуклеиновых кислот может использоваться функция "Blast 2 sequences" программы BLASTTM (Blastn) с использованием параметров настройки по умолчанию. Последовательности нуклеиновых кислот с еще большим подобием относительно референсных последовательностей показывают возрастающий процент идентичности при оценке с помощью данного метода.
Специфично губридизуемый/специфично комплементарный: При использовании в настоящем описании термины "специфично губридизуемый" и "специфично комплементарный" являются терминами, которые указывают на достаточную степень комплементарности, при которой между молекулой нуклеиновой кислоты и целевой молекулой нуклеиновой кислоты происходит стабильное и специфическое связывание. Гибридизация между двумя молекулами нуклеиновых кислот включает образование антипараллельного выравнивания между нуклеотидными последовательностями двух молекул нуклеиновых кислот. Эти две молекулы при этом способны образовывать водородные связи с соответствующими основаниями на противоположной цепи, с образованием двухцепочечной молекулы, которая, при условии своей достаточной стабильности, может быть обнаружена с использованием методов, известных в уровне техники. Молекула нуклеиновой кислоты не должна быть на 100% комплементарной своей целевой последовательности, чтобы являться специфично губридизуемой. Впрочем, величина комплементарности последовательности, которая должна существовать для специфичной гибридизации, зависит от используемых условий гибридизации.
Условия гибридизации, которые дают определенную степень строгости, изменяются в зависимости от природы выбранного метода гибридизации, а также от состава и длины гибридизуемых последовательностей нуклеиновых кислот. Как правило, строгость гибридизации определяет температура гибридизации и ионная сила (в особенности концентрация Na+ и/или Mg++) гибридизационного буфера, хотя на строгость также влияет длительность промывки. Вычисления по условиям гибридизации, требуемым для достижения определенной степени строгости, известны средним специалистам в данной области техники и обсуждаются, например, в Sambrook et al. (ed.) Molecular Cloning: A Laboratory Manual, 2nd ed., vol. 1-3, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, 1989, главы 9 и 11; и Hames and Higgins (eds.) Nucleic Acid Hybridization, IRL Press, Oxford, 1985. Более подробную инструкцию и руководство по гибридизации нуклеиновых кислот можно найти, например, в Tijssen, "Overview of principles of hybridization and the strategy of nucleic acid probe assays", в Laboratory Techniques in Biochemistry and Molecular Biology- Hybridization with Nucleic Acid Probes, Part I, Chapter 2, Elsevier, NY, 1993; и Ausubel et al., Eds., Current Protocols in Molecular Biology, Chapter 2, Greene Publishing and Wiley-Interscience, NY, 1995.
При использовании в настоящем описании, "строгие условия" охватывают условия, при которых гибридизация будет происходить только тогда, если между гибридизуемой молекулой и последовательностью в целевой молекуле нуклеиновой кислоты будет меньше 20% несоответствия. "Строгие условия" включают более конкретные уровни строгости. Таким образом, при использовании в настоящем описании, условия "умеренной строгости" являются такими условиями, при которых молекулы с более чем 20% несоответствием последовательности не будут гибридизоваться; условия "высокой строгости" являются такими условиями, при которых последовательности с более чем 10% несоответствием не будут гибридизоваться; и условия "очень высокой строгости" являются такими условиями, при которых последовательности с более чем 5% несоответствием не будут гибридизоваться. Следующее ниже является репрезентативными, неограничивающими условиями гибридизации.
Условие высокой строгости (позволяет обнаруживать последовательности, которые обладают по меньшей мере 90% идентичностью последовательности): Гибридизация в 5× буфере SSC (где буфер SSC содержит детергент, такой как SDS, а также дополнительные реактивы, такие как ДНК спермы лосося, ЭДТА и т.д.) при 65°C в течение 16 часов; две промывки в 2× буфере SSC (где буфер SSC содержит детергент, такой как SDS, а также дополнительные реактивы, такие как ДНК спермы лосося, ЭДТА и т.д.) при комнатной температуре, по 15 минут каждая; и две промывки в 0,5× буфере SSC (где буфер SSC содержит детергент, такой как SDS, а также дополнительные реактивы, такие как ДНК спермы лосося, ЭДТА и т.д.) при 65°C, по 20 минут каждая.
Условие умеренной строгости (позволяет обнаруживать последовательности, которые обладают по меньшей мере 80% идентичностью последовательности): Гибридизация в 5×-6× буфере SSC (где буфер SSC содержит детергент, такой как SDS, а также дополнительные реактивы, такие как ДНК спермы лосося, ЭДТА и т.д.) при 65-70°C в течение 16-20 часов; две промывки в 2× буфере SSC (где буфер SSC содержит детергент, такой как SDS, а также дополнительные реактивы, такие как ДНК спермы лосося, ЭДТА и т.д.) при комнатной температуре, по 5-20 минут каждая; и две промывки в 1× буфере SSC (где буфер SSC содержит детергент, такой как SDS, а также дополнительные реактивы, такие как ДНК спермы лосося, ЭДТА и т.д.) при 55-70°C, по 30 минут каждая.
Контрольное условие низкой строгости (гибридизуются последовательности, которые обладают по меньшей мере 50% идентичностью последовательности): Гибридизация в 6× буфере SSC (где буфер SSC содержит детергент, такой как SDS, а также дополнительные реактивы, такие как ДНК спермы лосося, ЭДТА и т.д.) при температуре от комнатной до 55°C в течение 16-20 часов; промывка по меньшей мере два раза в 2×-3× буфере SSC (где буфер SSC содержит детергент, такой как SDS, а также дополнительные реактивы, такие как ДНК спермы лосося, ЭДТА и т.д.) при температуре от комнатной до 55°C, по 20-30 минут каждая.
При использовании в настоящем описании, термин "по существу гомологичный" или "существенная гомология", в отношении непрерывной последовательности нуклеиновой кислоты, относится к непрерывным нуклеотидным последовательностям, которые гибридизуются в строгих условиях с референсной последовательностью нуклеиновой кислоты. Например, последовательности нуклеиновых кислот, которые являются по существу гомологичными референсной последовательности нуклеиновой кислоты, являются такими последовательностями нуклеиновых кислот, которые гибридизуются в строгих условиях (например, условиях умеренной строгости, указанных выше) с референсной последовательностью нуклеиновой кислоты. По существу гомологичные последовательности могут обладать по меньшей мере 80% идентичностью последовательности. Например, по существу гомологичные последовательности могут обладать приблизительно от 80% до 100% идентичности последовательности, например, приблизительно 81%; приблизительно 82%; приблизительно 83%; приблизительно 84%; приблизительно 85%; приблизительно 86%; приблизительно 87%; приблизительно 88%; приблизительно 89%; приблизительно 90%; приблизительно 91%; приблизительно 92%; приблизительно 93%; приблизительно 94% приблизительно 95%; приблизительно 96%; приблизительно 97%; приблизительно 98%; приблизительно 98,5%; приблизительно 99%; приблизительно 99,5% и приблизительно 100%. Свойство существенной гомологии тесно связано со специфичной гибридизацией. Например, молекула нуклеиновой кислоты способна к специфичной губридизации, когда присутствует достаточная степень комплементарности, позволяющая избежать неспецифичного связывания нуклеиновой кислоты с нецелевыми последовательностями при условиях, когда требуется специфичное связывание, например, при строгих условиях гибридизации.
В некоторых случаях "гомологичный" может использоваться для обозначения отношения первого гена ко второму гену по происхождению от общей предшествующей последовательности ДНК. В таких случаях термин гомолог указывает на отношение между генами, разделенными событием видообразования (см. ортолог), или на отношение между генами, разделенными событием генетической дупликации (см. паралог). В других случаях "гомологичный" может использоваться для обозначения уровня идентичности последовательностей между одной или более полинуклеотидными последовательностями, в таких случаях одна или более полинуклеотидных последовательностей не обязательно происходят от общей предшествующей последовательности ДНК. Специалисты в данной области техники осведомлены о взаимозаменяемости термина "гомологичный" и принимают во внимание надлежащее применение термина.
При использовании в настоящем описании, термин "ортолог" (или "ортологичный") относится к гену в двух или более биологических видах, который произошел от общей предшествующей нуклеотидной последовательности и может сохранять одну и ту же функцию в двух или более биологических видах.
При использовании в настоящем описании, термин "паралог" относится к генам, связанным дупликацией в геноме. Ортологи сохраняют одну и ту же функцию в ходе развития, тогда как паралоги приобретают новые функции, даже если эти новые функции не связаны с исходной функцией гена.
При использовании в настоящем описании, две молекулы последовательности нуклеиновых кислот, как говорят, демонстрируют "полную комплементарность", когда каждый нуклеотид последовательности, считываемой в 5'-3' направлении, комплементарен каждому нуклеотиду другой последовательности, считываемой в 3'-5' направлении. Нуклеотидная последовательность, которая комплементарна референсной нуклеотидной последовательности, будет демонстрировать последовательность, идентичную обратной комплементарной последовательности референсной нуклеотидной последовательности. Эти термины и описания хорошо определены в уровне техники и понятны средним специалистам в данной области.
При определении процента идентичности последовательности между аминокислотными последовательностями, специалистам в данной области известно, что идентичность аминокислоты в данном положении, обеспечиваемая выравниванием, может отличаться без влияния на желательные свойства полипептидов, составляющих выравниваемые последовательности. В этих случаях процент идентичности последовательности можно регулировать с учетом подобия между консервативно замененными аминокислотами. Такое регулирование известно и обычно используется специалистами в данной области. См., например, Myers and Miller (1988) Computer Applications in Biosciences 4:11-7. В уровне техники известны статистические методы, которые могут использоваться при анализе 7018 идентифицированных оптимальных геномных локусов.
В качестве варианта осуществления, идентифицированные оптимальные геномные локусы, включающие 7018 отдельных последовательностей оптимальных геномных локусов, могут быть проанализированы с помощью критерия F-распределения. В теории вероятности и статистике, F-распределение представляет собой непрерывное распределение вероятности. Критерий F-распределения представляет собой критерий статистической значимости, который имеет F-распределение, и используется при сравнении статистических моделей, которые были согласованы с набором данных, для идентификации оптимальной согласованной модели. F-распределение представляет собой непрерывное распределение вероятности, и также известно как F-распределение Снедекора или распределение Фишера-Снедекора. F-распределение часто возникает как нулевое распределение статистики критерия, наиболее характерное в дисперсионном анализе. F-распределение является скошенным вправо распределением. F-распределение является асимметричным распределением, которое имеет минимальное значение 0, но не имеет максимального значения. Кривая достигает пика справа, близко от 0, и затем постепенно приближается к горизонтальной оси с ростом F значения. F-распределение приближается, но никогда не достигает горизонтальной оси. Следует понимать, что в других вариантах осуществления вариации в этом уравнении, или даже разных уравнениях, могут быть получены и использованы специалистом, и могут быть применены для анализа 7018 отдельных последовательностей оптимальных геномных локусов.
Функционально связанный: первая нуклеотидная последовательность "функционально связана" со второй нуклеотидной последовательностью, когда первая нуклеотидная последовательность находится в функциональной зависимости со второй нуклеотидной последовательностью. Например, промотор функционально связан с кодирующей последовательностью, если промотор воздействует на транскрипцию или экспрессию кодирующей последовательности. В случае рекомбинантного получения, функционально связанные нуклеотидные последовательности обычно являются непрерывными и, в тех случаях, когда необходимо соединить две кодирующие белок области, находятся в одной рамке считывания. Впрочем, нуклеотидные последовательности не должны быть непрерывными, чтобы быть функционально связанными.
Термин "функционально связанный", при использовании в отношении регуляторной последовательности и кодирующей последовательности, означает, что регуляторная последовательность влияет на экспрессию связанной кодирующей последовательности. "Регуляторные последовательности", "регуляторные элементы" или "элементы контроля" относятся к нуклеотидным последовательностям, которые влияют на время и уровень/величину транскрипции, процессинга или стабильности РНК, или трансляции связанной кодирующей последовательности. Регуляторные последовательности могут включать промотры; лидерные последовательности трансляции; интроны; энхансеры; структуры стебель-петля; последовательности связывания репрессора; последовательности терминации; последовательности распознавания полиаденилирования и т.д. Определенные регуляторные последовательности могут быть расположены до и/или после кодирующей последовательности, функционально связанной с ними. Кроме того, определенные регуляторные последовательности, функционально связанные с кодирующей последовательностью, могут быть расположены на связанной комплементарной цепи двухцепочечной молекулы нуклеиновой кислоты.
При использовании в отношении двух или более аминокислотных последовательностей, термин "функционально связанный" означает, что первая аминокислотная последовательность находится в функциональной взаимосвязи по меньшей мере с одной из дополнительных аминокислотных последовательностей.
Раскрытые способы и композиции включают слитые белки, включающие расщепляющий домен, функционально связанный с ДНК-связывающим доменом (например, ZFP), где ДНК-связывающий домен при связывании с последовательностью в оптимальном геномном локусе сои направляет активность расщепляющего домена вблизи последовательности и, следовательно, вызывает разрыв двойной цепи в оптимальном геномном локусе. Как указано в других частях настоящего описания, цинк-пальцевый домен может быть сконструирован для связывания фактически с любой нужной последовательностью. Таким образом, один или более ДНК-связывающих доменов могут быть сконструированы для связывания с одной или более последовательностями в оптимальном геномном локусе. Экспрессия слитого белка, включающего ДНК-связывающий домен и расщепляющий домен, в клетке вызывает расщепление в или вблизи целевого участка.
ВАРИАНТЫ ОСУЩЕСТВЛЕНИЯ
Направление трансгенов и пакетов трансгенов в определенные положения в геноме двудольных растений, таких как растение сои, будет улучшать качество трансгенных объектов, снижать затраты, связанные с получением трансгенных объектов, и обеспечит новые способы получения трансгенных растительных продуктов, такие как последовательный стэкинг генов. В целом, направление трансгенов в определенные участки генома, вероятно, будет коммерчески выгодным. Значительные успехи были достигнуты в последние несколько лет в направлении разработки сайт-специфических нуклеаз, таких как ZFN, CRISPR и TALENS, которые могут способствовать введению донорных полинуклеотидов в предварительно выбранные участки в растительных и других геномах. Однако намного меньше известно о признаках геномных участков, которые подходят для таргетинга. Исторически сложилось, что несущественные гены и участки интеграции патогенов (вирусов) в геномах использовались в качестве локусов для таргетинга. Количество таких участков в геномах скорее является лимитирующим, и поэтому существует потребность в идентификации и исследовании оптимальных геномных локусов-мишеней, которые могут использоваться для таргетинга донорных полинуклеотидных последовательностей. В дополнение к возможности таргетинга, оптимальные геномные локусы, как ожидают, будут нейтральными участками, которые смогут поддерживать экспрессию трансгена и применение в селекции.
Заявители выяснили, что для участков вставки требуются дополнительные критерии, и объединили эти критерии, чтобы идентифицировать и выбрать оптимальные участки в геноме двудольного растения, таком как геном сои, для вставки экзогенных последовательностей. В целях таргетинга участок выбранной вставки должен быть уникальным и находиться в не содержащей повторы области генома двудольного растения, такого как растение сои. Аналогичным образом, оптимальный геномный участок для вставки должен оказывать минимальные нежелательные фенотипические воздействия и являться чувствительным к событиям рекомбинации, чтобы облегчить интрогрессию в агрономически элитные линии с использованием традиционных методик селекции. Чтобы идентифицировать геномные локусы, которые соответствуют перечисленным критериям, геном растения сои был исследован с использованием специализированного биоинформационного метода и масштабных наборов геномных данных для идентификации новых геномных локусов, обладающих особенностями, которые полезны для интеграции донорной полинуклеотидной последовательности и последующей экспрессии вставленной кодирующей последовательности.
I. Идентификация негенных геномных локусов сои
В соответствии с одним вариантом осуществления предложен способ идентификации оптимальной негенной геномной последовательности сои для вставки экзогенных последовательностей. Способ включает этапы вначале идентификации геномных последовательностей сои длиной по меньшей мере 1 тпн, которые являются гипометилированными. В одном варианте осуществления гипометилированная геномная последовательность имеет длину 1, 1,5, 2, 2,5, 3, 3,5, 4, 4,5, 5, 5,5, 6, 6,5, 7, 7,5, 8, 8,5, 9, 10, 11, 12, 13, 14, 15, 16 или 17 тпн. В одном варианте осуществления гипометилированная геномная последовательность имеет длину от приблизительно 1 до приблизительно 5,7 тпн, а в другом варианте осуществления она имеет длину приблизительно 2 тпн. Последовательность считается гипометилированной, если она имеет менее 1% метилирования ДНК в последовательности. В одном варианте осуществления статус метилирования измеряют по присутствию 5-метилцитозина в одном или более CpG динуклеотидах, CHG или CHH тринуклеотидах в выбранной последовательности сои, по отношению к общему количеству цитозинов, присутствующих в соответствующих CpG динуклеотидах, CHG или CHH тринуклеотидах в нормальном контрольном образце ДНК. На метилирование CHH указывает 5-метилцитозин, после которого следуют два нуклеотида, которые могут не быть гуанином, и метилирование CHG относится к присутствию 5-метилцитозина перед основаниями аденином, тимином или цитозином, после которых следует гуанин. Более конкретно, в одном варианте осуществления выбранная последовательность сои содержит менее 1, 2 или 3 метилированных нуклеотидов на 500 нуклеотидов выбранной последовательности сои. В одном варианте осуществления выбранная последовательность сои содержит менее одного, двух или трех 5-метилцитозинов в CpG динуклеотидах на 500 нуклеотидов выбранной последовательности сои. В одном варианте осуществления выбранная последовательность сои имеет длину 1-4 тпн и включает 1 тпн последовательность, не содержащую 5-метилцитозинов. В одном варианте осуществления выбранная последовательность сои имеет длину 1, 1,5, 2, 2,5, 3, 3,5, 4, 4,5, 5, 5,5 или 6 тпн и содержит 1 или 0 метилированных нуклеотидов на протяжении всей своей длины. В одном варианте осуществления выбранная последовательность сои имеет длину 1, 1,5, 2, 2,5, 3, 3,5, 4, 4,5, 5, 5,5 или 6 тпн и не содержит 5-метилцитозинов в CpG динуклеотидах на протяжении всей своей длины. В соответствии с одним вариантом осуществления метилирование выбранной последовательности сои может изменяться в зависимости от исходной ткани. В таких вариантах осуществления уровни метилирования, используемые для определения, является ли последовательность гипометилированной, представляют собой среднее количество метилирования в последовательностях, выделенных из двух или более тканей (например, из корня и побега).
В дополнение к требованию, чтобы оптимальный геномный участок являлся гипометилированным, выбранная последовательность сои также должна быть негенной. Таким образом, все гипометилированные геномные последовательности также подвергают скринингу с целью исключения гипометилированных последовательностей, которые содержат генную область. Это включает любые открытые рамки считывания независимо от того, кодирует ли транскрипт белок. Гипометилированные геномные последовательности, которые включают генные области, включающие любые идентифицируемые смежные 5′ и 3′ некодирующие нуклеотидные последовательности, участвующие в регуляции экспрессии открытой рамки считывания, и любые интроны, которые могут присутствовать в генной области, исключают из оптимального негенного геномного локуса сои согласно настоящему описанию.
Оптимальные негенные геномные локусы сои также должны являться последовательностями, которые продемонстрировали подтверждение рекомбинации. В одном варианте осуществления выбранная последовательность сои должна быть такой последовательностью, в которой обнаружили по меньшей мере одно событие рекомбинации между двумя маркерами, фланкирующими выбранную последовательность сои, как обнаружено с использованием набора данных маркеров высокого разрешения, полученного из множества картирующих популяций. В одном варианте осуществления пара маркеров, фланкирующих 0,5, 1, 1,5 мпн геномную последовательность двудольного растения, такую как геномную последовательность сои, включающую выбранную последовательность сои, используют для вычисления рекомбинантной частоты для выбранной последовательности сои. Отношение частот рекомбинации между каждой парой маркеров (измеряемых в сантиморганах (сМ)) к геномному физическому расстоянию между маркерами (в мпн)) должно быть больше 0,0157 сМ/мпн. В одном варианте осуществления частота рекомбинации для 1 мпн геномной последовательности сои, включающей выбранную последовательность сои, изменяется в пределах от приблизительно 0,01574 сМ/мпн до приблизительно 83,52 сМ/мпн. В одном варианте осуществления оптимальные геномные локусы являются такими локусами, в которых события рекомбинации были обнаружены в пределах выбранной последовательности сои.
Оптимальные негенные геномные локусы сои также будут являться последовательностью-мишенью, то есть последовательностью, которая является относительно уникальной в геноме сои, при этом ген, направляемый в выбранную последовательность сои, будет встраиваться только в одно положение генома сои. В одном варианте осуществления вся длина оптимальной геномной последовательности обладает меньше чем 30%, 35% или 40% идентичностью последовательности с другой последовательностью подобной длины, содержащейся в геноме сои. Таким образом, в одном варианте осуществления выбранная последовательность сои не может включать 1 тпн последовательность, которая обладает более чем 25%, 30%, 35% или 40% идентичностью последовательности с другой 1 тпн последовательностью, содержащейся в геноме сои. В другом варианте осуществления выбранная последовательность сои не может включать 500 пн последовательность, которая обладает более чем 30%, 35% или 40% идентичностью последовательности с другой 500 пн последовательностью, содержащейся в геноме сои. В одном варианте осуществления выбранная последовательность сои не может включать 1 тпн последовательность, которая обладает более чем 40% идентичностью последовательности с другой 1 тпн последовательностью, содержащейся в геноме двудольного растения, такого как растение сои.
Оптимальные негенные геномные локусы сои также будут расположены проксимально относительно генной области. Более конкретно, выбранная последовательность сои должна быть расположена вблизи генной области (например, генная область должна быть расположена в пределах 40 тпн геномной последовательности, фланкирующей и примыкающей к любому концу выбранной последовательности сои, присутствующей в нативном геноме). В одном варианте осуществления генная область расположена в пределах 10, 20, 30 или 40 тпн примыкающей геномной последовательности, фланкирующей любой конец выбранной последовательности сои, присутствующей в нативном геноме сои. В одном варианте осуществления две или больше генных области расположены в пределах 10, 20, 30 или 40 тпн примыкающей геномной последовательности, фланкирующей два конца выбранной последовательности сои. В одном варианте осуществления 1-18 генных областей расположены в пределах 10, 20, 30 или 40 тпн примыкающей геномной последовательности, фланкирующей два конца выбранной последовательности сои. В одном варианте осуществления две или более генных областей расположены в пределах 20, 30 или 40 тпн геномной последовательности, включающей выбранную последовательность сои. В одном варианте осуществления 1-18 генных областей расположены в пределах 40 тпн геномной последовательности, включающей выбранную последовательность сои. В одном варианте осуществления генная область, расположенная в пределах 10, 20, 30 или 40 тпн примыкающей геномной последовательности, фланкирующей выбранную последовательность сои, включает известный ген в геноме двудольного растения, такого как растение сои.
В соответствии с одним вариантом осуществления предложены измененные негенные геномные локусы сои, где локусы имеют длину по меньшей мере 1 тпн, являются негенными, не содержат метилированных остатков цитозина, имеют частоту рекомбинации больше 0,01574 сМ/мпн на протяжении 1 мпн геномной области, охватывающей геномные локусы сои, и 1 тпн последовательность геномных локусов сои обладает менее чем 40% идентичностью последовательности с любой другой 1 тпн последовательностью, содержащейся в геноме двудольного растения, где негенные геномные локусы сои изменены посредством вставки целевой ДНК в негенные геномные локусы сои.
В соответствии с одним вариантом осуществления предложен способ идентификации оптимальных негенных геномных локусов двудольного растения, включающих, например, геномные локусы сои. В некоторых вариантах осуществления способ сначала включает скрининг генома двудольного растения для создания первого пула выбранных последовательностей сои, которые имеют минимальную длину 1 тпн и являются гипометилированными, где геномная последовательность необязательно имеет меньше 1% метилирования, где геномная последовательность необязательно не содержит метилированных остатков цитозина. Указанный первый пул выбранных последовательностей сои может быть затем подвергнут скринингу с целью исключения локусов, которые не отвечают требованиям в отношении оптимальных негенных геномных локусов сои. Геномные последовательности двудольного растения, такие как геномные последовательности, полученные из сои, которые кодируют транскрипты двудольного растения, обладают более чем 40% или более высокой идентичностью последовательности с другой последовательностью подобной длины, не демонстрируют подтверждение рекомбинации и не имеют известной открытой рамки считывания в пределах 40 тпн выбранной последовательности сои, исключают из первого пула последовательностей с получением второго пула последовательностей, которые относятся к оптимальным негенным локусам сои. В одном варианте осуществления любые выбранные последовательности сои, которые не имеют известного гена двудольного растения (то есть гена сои), или последовательность, включающая 2 тпн область до и/или 1 тпн область после известного гена двудольного растения, в пределах 40 тпн от одного конца указанной негенной последовательности, исключаются из первого пула последовательностей. В одном варианте осуществления исключают любые выбранные последовательности сои, которые не содержат известный ген, который экспрессирует белок, в пределах 40 тпн выбранной последовательности сои. В одном варианте осуществления исключают любые выбранные последовательности сои, которые не имеют частоту рекомбинации больше 0,01574 сМ/мпн.
Применяя указанные критерии отбора, заявители идентифицировали отобранные оптимальные геномные локусы двудольного растения, такого как сои, которые служат в качестве оптимальных негенных геномных локусов сои, последовательности которых раскрыты в SEQ ID NO: 1-SEQ ID NO: 7018. Настоящее описание также охватывает природные варианты или модифицированные производные идентифицированных оптимальных негенных геномных локусов сои, где варианты или производные локусов включают последовательность, которая отличается от любой последовательности в SEQ ID NO: 1-SEQ ID NO: 7018 на 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 нуклеотидов. В одном варианте осуществления оптимальные негенные геномные локусы сои для применения в соответствии с настоящим описанием включают последовательности, выбранные из SEQ ID NO: 1-SEQ ID NO: 7018, или последовательности, которые обладают 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичности последовательности с последовательностью, выбранной из SEQ ID NO: 1-SEQ ID NO: 7018.
В другом варианте осуществления двудольные растения для применения в соответствии с настоящим описанием включают любое растение, выбранное из группы, состоящей из растения сои, растения канолы, растения рапса, растения Brassica, растения хлопка и растения подсолнечника. Примеры двудольных растений, которые могут применяться, включают, без ограничения перечисленными, канолу, хлопок, картофель, киноа, амарант, гречиху, сафлор, сою, сахарную свеклу, подсолнечник, канолу, рапс, табак, Arabidopsis, Brassica и хлопок.
В другом варианте осуществления оптимальные негенные геномные локусы сои для применения в соответствии с настоящим описанием включают последовательности, выбранные из растений сои. В другом варианте осуществления оптимальные негенные геномные локусы сои для применения в соответствии с настоящим описанием включают последовательности, выбранные из инбредных растений Glycine max. Таким образом, инбредное растение Glycine max включает соответствующие агрономически элитные сорта. В следующем варианте осуществления оптимальные негенные геномные локусы сои для применения в соответствии с настоящим описанием включают последовательности, выбранные из трансформируемых линий сои. В варианте осуществления репрезентативные трансформируемые линии сои включают; Maverick, Williams82, Merrill JackPeking, Suzuyutaka, Fayette, Enrei, Mikawashima, WaseMidori, Jack, Leculus, Morocco, Serena, Maple prest, Thorne, Bert, Jungery, A3237, Williams, Williams79, AC Colibri, Hefeng 25, Dongnong 42, Hienong 37, Jilin 39, Jiyu 58, A3237, Kentucky Wonder, Minidoka и их производные. Специалисту в данной области техники будет очевидно, что в результате филогенетической дивергенции различные типы линий сои не содержат идентичные геномные последовательности ДНК, и что в геномных последовательностях могут присутствовать полиморфизмы или аллельные вариации. В варианте осуществления настоящее описание охватывает такие полиморфизмы или аллельные вариации идентифицированных оптимальных негенных геномных локусов сои, где полиморфизмы или аллельные вариации включают последовательность, которая отличается от любой последовательности в SEQ ID NO: 1-SEQ ID NO: 7018 на 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 нуклеотидов. В другом варианте осуществления настоящее описание охватывает такие полиморфизмы или аллельные вариации идентифицированных оптимальных негенных геномных локусов сои, где последовательности, включающие полиморфизмы или аллельные вариации, обладают 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичностью последовательности с любой последовательностью SEQ ID NO: 1-SEQ ID NO: 7018.
Идентифицированные оптимальные геномные локусы, включающие 7018 индивидуальных последовательностей, могут быть распределены в различные подгруппы при последующем анализе с использованием метода многофакторного анализа. Применение любых статистических программ многофакторного анализа используется для выявления скрытой структуры (величин) множества переменных. Можно использовать много различных типов многофакторных алгоритмов, например, набор данных может быть проанализирован с использованием множественного регрессионного анализа, логистического регрессионного анализа, дискриминантного анализа, многофакторного дисперсионного анализа (MANOVA), факторного анализа (включающего простой факторный анализ и анализ главных компонент), кластерного анализа, многомерного шкалирования, анализа соответствий, анализа совмещения, канонического анализа, канонической корреляции и моделирования структурными уравнениями.
В соответствии с одним вариантом осуществления оптимальные негенные геномные локусы сои также анализируют с использованием многофакторного анализа данных, такого как анализ главных компонент (PCA). Здесь будет приведено только краткое описание, дополнительную информацию можно найти в H. Martens, T. Naes, Multivariate Calibration, Wiley, N.Y., 1989. Метод PCA позволяет оценить основную размерность (скрытые переменные) данных и дает обзор доминантных структур и основных тенденций в данных. В одном варианте осуществления оптимальные негенные геномные локусы сои могут быть отсортированы в кластеры с помощью статистического метода анализа главных компонент (PCA). Метод PCA представляет собой математическую процедуру, в которой используется ортогональное преобразование для превращения множества наблюдений вероятно коррелированных переменными в набор значений линейно некоррелированных переменных, названных главными компонентами. Количество главных компонент меньше или равно количеству исходных переменных. Это преобразование определяют таким способом, что первая главная компонента имеет наибольшую дисперсию (то есть дает максимально возможную вариабельность данных), при этом каждая последующая компонента в свою очередь имеет наибольшую дисперсию, возможную при ограничении, что она ортогональна (то есть некоррелирует) предыдущим компонентам. Главные компоненты должны быть независимыми, если набор данных совместно нормально распределен. Метод PCA чувствителен к относительному пропорциональному изменению исходных переменных. Примеры использования PCA для кластеризации множества объектов на основе характеристик объектов, включают: Ciampitti, I. et al., (2012) Crop Science, 52(6); 2728-2742, Chemometrics: A Practical Guide, Kenneth R. Beebe, Randy J. Pell, and Mary Beth Seasholtz, Wiley-Interscience, 1 edition, 1998, патент США 8,385,662 и европейский патент 2,340,975.
В соответствии с одним вариантом осуществления анализ главных компонент (PCA) проводили на 7018 оптимальных геномных локусах сои при использовании следующих 10 характеристик каждого идентифицированного оптимального геномного локуса сои:
1. Длина гипометилированной области вокруг оптимальных геномных локусов сои (OGL)
a. Профили метилирования ДНК в тканях корней и побегов, выделенных из двудольного растения, например, Glycine Max сорта Williams82, создавали с использованием метода высокопроизводительного полногеномного секвенирования. Выдеденную ДНК подвергали обработке бисульфитом, в результате которой неметилированные цитозины превращаются в урацилы, при этом на метилированные цитозины бисульфит не воздействует, и затем секвенировали с использованием технологии Illumina HiSeq (Krueger, F. et al. DNA methylome analysis using short bisulfite sequencing data. Nature Methods 9, 145–151 (2012)). Необработанные сиквенсы картировали с референсной последовательностью двудольного растения, например, референсной последовательностью Glycine max, при использовании программы картирования BismarkTM (как описано в Krueger F, Andrews SR (2011) Bismark: a flexible aligner and methylation caller for Bisulfite-Seq applications. (Bioinformatics 27: 1571–1572)). Длину гипометилированной области вокруг каждого OGL вычисляли при использовании описанных профилей метилирования.
2. Частота рекомбинации в 1 мпн области вокруг OGL
a. Для каждого OGL идентифицировали пару маркеров по обеим сторонам OGL, в окне 1 мпн. Частоту рекомбинации между каждой парой маркеров на хромосоме вычисляли на основе отношения генетического расстояния между маркерами (в сантиморганах (сМ)) к геномному физическому расстоянию между маркерами (в мпн).
3. Уровень уникальности последовательности OGL
a. Для каждого OGL нуклеотидную последовательность OGL сканировали по геному двудольного растения, например, геному сои сорта Williams82, с использованием поиска гомологии на основе BLAST. Поскольку эти последовательности OGL идентифицированы из генома двудольного растения, например, генома сои сорта Williams82, первое совпадение в BLAST, идентифицированное с помощью такого поиска, представляет собой саму последовательность OGL. Второе совпадение в BLAST идентифицировали для каждого OGL и охват выравнивания данного совпадения использовали в качестве степени уникальности последовательности OGL в геноме двудольного растения, например, геноме сои.
4. Расстояние от OGL до ближайшего соседнего гена
a. Информацию по аннотированию генов и местоположению известных генов в геноме двудольного растения, например, геноме сои сорта Williams82, получали из известной базы данных генома двудольного растения, например, базы данных генома сои (www.soybase.org). Для каждого OGL идентифицировали ближайший аннотированный ген до или после него, а также измеряли расстояние между последовательностью OGL и геном (в пн).
5. % GC в окружении OGL
a. Для каждого OGL анализировали нуклеотидную последовательность, чтобы оценить количество присутствующих оснований гуанина и цитозина. Этот показатель был представлен как процент от длины последовательности каждого OGL и дает значение % GC.
6. Количество генов в 40 тпн области вокруг OGL
a. Информацию по аннотированию генов и местоположение известных генов в геноме двудольного растения, например, геноме сои сорта Williams82, получали из известной базы данных генома двудольного растения, например, базы данных генома сои (www.soybase.org). Для каждого OGL определяли 40 тпн окно вокруг OGL и подсчитывали количество аннотированных генов, местоположения которых перекрывались с этим окном.
7. Средняя экспрессия генов в 40 тпн области вокруг OGL.
a. Уровень экспрессии транскриптов генов двудольного растения, например генов сои, измеряли с помощью анализа данных профилирования транскриптомов, полученных из тканей двудольного растения, например тканей корней и побегов сои сорта Williams82, при использовании технологии RNAseq. Для каждого OGL идентифицировали аннотированные гены в геноме двудольного растения, геноме сои сорта Williams82, которые присутствовали в 40 тпн области вокруг OGL. Уровни экспрессии для каждого гена в окне получали из транскриптомных профилей и вычисляли средний уровень экспрессии.
8. Уровень занятости нуклеосомами вокруг OGL
a. Установление уровня занятости нуклеосомами для определенной нуклеотидной последовательности дает информацию о функциях хромосомы и геномном окружении последовательности. Статистический пакет NuPoPTM обеспечивает легкий в применении программный инструмент для предсказания занятости нуклеосомами и построения карты наиболее вероятного расположения нуклеосом для геномных последовательностей любого размера (Xi, L., Fondufe-Mittendor, Y., Xia, L., Flatow, J., Widom, J. and Wang, J.-P., Predicting nucleosome positioning using a duration Hidden Markov Model, BMC Bioinformatics, 2010, doi:10.1186/1471-2105-11-346). Для каждого OGL нуклеотидную последовательность вводили в программу NuPoPTM и вычисляли показатель занятости нуклеосомами.
9. Относительное местоположение на хромосоме (близость к центромере)
a. Информацию о положении центромеры в каждой из хромосом двудольного растения, например, хромосом сои, и длину плечей хромосом получали из базы данных генома двудольного растения, например, базы данных генома сои (www.soybase.org). Для каждого OGL измеряли геномное расстояние (в пн) от последовательности OGL до центромеры хромосомы, на которой он расположен. Относительное местоположение OGL на хромосоме представлено как отношение ее геномного расстояния до центромеры к длине плеча конкретной хромосомы, на которой он находится.
10. Количество OGL в 1 мпн области вокруг OGL
a. Для каждого OGL определяли геномное окно протяженностью 1 мпн вокруг местоположения OGL и подсчитывали количество OGL в наборе данных 1 тпн OGL двудольного растения, геномные местоположения которых перекрывались с этим окном.
Результаты или значения для показателя характеристик и свойств каждого оптимального негенного геномного локуса сои описаны ниже в Таблице 3 Примера 2. Полученный в результате набор данных использовали в статистическом методе PCA для распределения 7018 идентифицированных оптимальных негенных геномных локусов сои в кластеры. Во время процесса кластеризации, после оценки "p" главных компонент оптимальных геномных локусов, распределение оптимальных геномных локусов в один из 32 кластеров продолжали в "p"-мерном евклидовом пространстве. Каждую из осей "p" разбивали на "k" интервалов. Оптимальные геномные локусы, распределенные в один и тот же интервал, группировали с формированием кластеров. Используя данный анализ, каждую ось PCA разбивали на два интервала, который выбирали на основе априорной информации относительно количества кластеров, требуемых для экспериментальной проверки. Весь анализ и визуализацию полученных кластеров выполняли с помощью программы Molecular Operating Environment™ (MOE), выпускаемой Chemical Computing Group Inc. (Montreal, Quebec, Canada). Метод PCA использовали для разделения набора из 7018 оптимальных геномных локусов сои на 32 различных кластера, исходя из их параметров, описанных выше.
Во время процесса PCA генерировали пять главных компонент (ГК), причем лучшие три ГК содержали приблизительно 90% общей вариации в наборе данных (Таблица 4). Эти три ГК использовали для графического представления 32 кластеров на трехмерном графике (см. Фиг. 1). После завершения процесса кластеризации из каждого кластера выбрали по одному репрезентативному оптимальному геномному локусу. Это было выполнено посредством выбора в каждом кластере отобранного оптимального геномного локуса, который был наиболее близок к центроиду данного кластера, с применением вычислительных методов (Таблица 4). Хромосомные местоположения 32 репрезентативных оптимальных геномных локусов однородно распределены по хромосомам сои, как показано на Фиг. 2.
В варианте осуществления предложена выделенная или очищенная последовательность оптимальных негенных геномных локусов сои, выбранная из любого кластера, описанного в Таблице 6 Примера 2. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 1. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 2. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 3. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 4. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 5. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 6. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 7. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 8. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 9. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 10. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 11. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 12. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 13. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 14. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 15. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 16. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 17. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 18. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 19. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 20. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 21. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 22. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 23. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 24. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 25. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 26. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 27. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 28. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 29. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 30. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 31. В одном из вариантов осуществления выделенная или очищенная последовательность оптимальных негенных геномных локусов сои является геномной последовательностью, выбранной из кластера 32.
В соответствии с одним из вариантов осуществления предложены модифицированные оптимальные негенные геномные локусы сои, где оптимальные негенные геномные локусы сои были модифицированы и включают замену, делецию или вставку одного или более нуклеотидов. В одном из вариантов осуществления оптимальные негенные геномные локусы сои модифицированы посредством вставки целевой ДНК, необязательно сопровождаемой последующими нуклеотидными дупликациями, делециями или инверсиями последовательности геномных локусов.
В варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из любого кластера, описанного в Таблице 6 Примера 2. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 2. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 3. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 4. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 5. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 6. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 7. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 8. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 9. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 10. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 11. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 12. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 13. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 14. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 15. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 16. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 17. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 18. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 19. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 20. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 21. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 22. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 23. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 24. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 25. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 26. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 27. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 28. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 29. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 30. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 31. В одном из вариантов осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 32.
В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30 или 31. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28 или 29. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27 или 28. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26 или 27. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25 или 26. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23 или 24. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22 или 23. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21 или 22. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20 или 21. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18 или 19. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17 или 18. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16 или 17. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 или 16. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13 или 14. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12 или 13. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или 11. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8 или 9. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7 или 8. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6 или 7. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5 или 6. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4 или 5. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3 или 4. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2 или 3. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1 или 2.
В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30 или 32.
В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 32.
В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28 или 32.
В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, или 27. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 9, 10, 11, 12, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 15, 16, 17, 18, 25, 26, 27, 28, 29, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 21, 22, 23, 24, 30, 31 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30 или 32. В другом варианте осуществления модифицируемые оптимальные негенные геномные локусы сои являются геномной последовательностью, выбранной из кластера 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29 или 31.
В одном варианте осуществления оптимальные негенные геномные локусы сои выбраны из геномных последовательностей soy_ogl_2474 (SEQ ID NO: 1), soy_ogl_768 (SEQ ID NO: 506), soy_ogl_2063 (SEQ ID NO: 2063), soy_ogl_1906 (SEQ ID NO: 1029), soy_ogl_1112 (SEQ ID NO: 1112), soy_ogl_3574 (SEQ ID NO: 1452), soy_ogl_2581 (SEQ ID NO: 1662), soy_ogl_3481 (SEQ ID NO: 1869), soy_ogl_1016 (SEQ ID NO: 2071), soy_ogl_937 (SEQ ID NO: 2481), soy_ogl_6684 (SEQ ID NO: 2614), soy_ogl_6801 (SEQ ID NO: 2874), soy_ogl_6636 (SEQ ID NO: 2970), soy_ogl_4665 (SEQ ID NO: 3508), soy_ogl_3399 (SEQ ID NO: 3676), soy_ogl_4222 (SEQ ID NO: 3993), soy_ogl_2543 (SEQ ID NO: 4050), soy_ogl_275 (SEQ ID NO: 4106), soy_ogl_598 (SEQ ID NO: 4496), soy_ogl_1894 (SEQ ID NO: 4622), soy_ogl_5454 (SEQ ID NO: 4875), soy_ogl_6838 (SEQ ID NO: 4888), soy_ogl_4779 (SEQ ID NO: 5063), soy_ogl_3333 (SEQ ID NO: 5122), soy_ogl_2546 (SEQ ID NO: 5520), soy_ogl_796 (SEQ ID NO: 5687), soy_ogl_873 (SEQ ID NO: 6087), soy_ogl_5475 (SEQ ID NO: 6321), soy_ogl_2115 (SEQ ID NO: 6520), soy_ogl_2518 (SEQ ID NO: 6574), soy_ogl_5551 (SEQ ID NO: 6775) и soy_ogl_4563 (SEQ ID NO: 6859).
В одном варианте осуществления оптимальные негенные геномные локусы сои выбраны из геномных последовательностей soy_ogl_308 (SEQ ID NO: 43), soy_ogl_307 (SEQ ID NO: 566), soy_ogl_2063 (SEQ ID NO: 748), soy_ogl_1906 (SEQ ID NO: 1029), soy_ogl_262 (SEQ ID NO: 1376), soy_ogl_5227 (SEQ ID NO: 1461), soy_ogl_4074 (SEQ ID NO: 1867), soy_ogl_3481 (SEQ ID NO: 1869), soy_ogl_1016 (SEQ ID NO: 2071), soy_ogl_937 (SEQ ID NO: 2481), soy_ogl_5109 (SEQ ID NO: 2639), soy_ogl_6801 (SEQ ID NO: 2874), soy_ogl_6636 (SEQ ID NO: 2970), soy_ogl_4665 (SEQ ID NO: 3508), soy_ogl_6189 (SEQ ID NO: 3682), soy_ogl_4222 (SEQ ID NO: 3993), soy_ogl_2543 (SEQ ID NO: 4050), soy_ogl_310 (SEQ ID NO: 4326), soy_ogl_2353 (SEQ ID NO: 4593), soy_ogl_1894 (SEQ ID NO: 4622), soy_ogl_3669 (SEQ ID NO: 4879), soy_ogl_3218 (SEQ ID NO: 4932), soy_ogl_5689 (SEQ ID NO: 5102), soy_ogl_3333 (SEQ ID NO: 5122), soy_ogl_2546 (SEQ ID NO: 5520), soy_ogl_1208 (SEQ ID NO: 5698), soy_ogl_873 (SEQ ID NO: 6087), soy_ogl_5957 (SEQ ID NO: 6515), soy_ogl_4846 (SEQ ID NO: 6571), soy_ogl_3818 (SEQ ID NO: 6586), soy_ogl_5551 (SEQ ID NO: 6775), soy_ogl_7 (SEQ ID NO: 6935), soy_OGL_684 (SEQ ID NO: 47), soy_OGL_682 (SEQ ID NO: 2101), soy_OGL_685 (SEQ ID NO: 48), soy_OGL_1423 (SEQ ID NO: 639), soy_OGL_1434 (SEQ ID NO: 137), soy_OGL_4625 (SEQ ID NO: 76) и soy_OGL_6362 (SEQ ID NO: 440).
В одном варианте осуществления в оптимальные негенные геномные локусы сои направляют целевую ДНК, где целевая ДНК интегрируется в или близко к сайтам-мишеням цинк-пальцевой нуклеазы. В соответствии с вариантом осуществления примеры сайтов-мишеней цинковых пальцев оптимальных отобранных геномных локусов кукурузы представлены в Таблице 8. В соответствии с вариантом осуществления интеграция целевой ДНК проходит в или близко к примерным сайтам-мишеням в: SEQ ID NO: 7363 и SEQ ID NO: 7364, SEQ ID NO: 7365 и SEQ ID NO: 7366, SEQ ID NO: 7367 и SEQ ID NO: 7368, SEQ ID NO: 7369 и SEQ ID NO: 7370, SEQ ID NO: 7371 и SEQ ID NO: 7372, SEQ ID NO: 7373 и SEQ ID NO: 7374, SEQ ID NO: 7375 и SEQ ID NO: 7376, SEQ ID NO: 7377 и SEQ ID NO: 7378, SEQ ID NO: 7379 и SEQ ID NO: 7380, SEQ ID NO: 7381 и SEQ ID NO: 7382, SEQ ID NO: 7383 и SEQ ID NO: 7384, SEQ ID NO: 7385 и SEQ ID NO: 7386, SEQ ID NO: 7387 и SEQ ID NO: 7388, SEQ ID NO: 7389 и SEQ ID NO: 7390, SEQ ID NO: 7391 и SEQ ID NO: 7392, SEQ ID NO: 7393 и SEQ ID NO: 7394, SEQ ID NO: 7395 и SEQ ID NO: 7396, SEQ ID NO: 7397 и SEQ ID NO: 7398, SEQ ID NO: 7399 и SEQ ID NO: 7400, SEQ ID NO: 7401 и SEQ ID NO: 7402, SEQ ID NO: 7403 и SEQ ID NO: 7404, SEQ ID NO: 7405 и SEQ ID NO: 7406, SEQ ID NO: 7407 и SEQ ID NO: 7408, SEQ ID NO: 7409 и SEQ ID NO: 7410, SEQ ID NO: 7411 и SEQ ID NO: 7412, SEQ ID NO: 7413 и SEQ ID NO: 7414, SEQ ID NO: 7415 и SEQ ID NO: 7416, SEQ ID NO: 7417 и SEQ ID NO: 7418, SEQ ID NO: 7419 и SEQ ID NO: 7420, SEQ ID NO: 7421 и SEQ ID NO: 7422, SEQ ID NO: 7423 и SEQ ID NO: 7424, SEQ ID NO: 7425 и SEQ ID NO: 7426.
В соответствии с вариантом осуществления цинк-пальцевая нуклеаза связывается с сайтом-мишенью цинкового пальца и расщепляет уникальные полинуклеотидные сайты-мишени в геноме сои, после чего целевая ДНК интегрируется по или близко к полинуклеотидным сайтам-мишеням в геном сои. В варианте осуществления интеграция целевой ДНК в сайт-мишень цинкового пальца может приводить к перестройкам. В соответствии с одним вариантом осуществления перестройки могут включать делеции, вставки, инверсии и повторы. В варианте осуществления интеграция целевой ДНК происходит близко к сайту-мишени цинкового пальца. Согласно аспекту варианта осуществления интеграция ДНК происходит близко к сайту-мишени цинкового пальца, при этом ДНК может интегрироваться в пределах 2 тпн, 1,75 тпн, 1,5 тпн, 1,25 тпн, 1,0 тпн, 0,75 тпн, 0,5 тпн или 0,25 тпн от сайта-мишени цинкового пальца. Вставка в геномной области, близко к сайту-мишени цинкового пальца, известна в уровне техники, см. патентную публикацию США 2010/0257638 A1 (полностью включенную в настоящую заявку посредстсвом отсылки).
В соответствии с одним вариантом осуществления выбранная негенная последовательность включает следующие характеристики:
a) негенная последовательность не содержит более чем 1% метилирования ДНК в последовательности;
b) негенная последовательность имеет относительное значение местоположения от 0,211 до 0,976 отношения геномного расстояния от центромеры хромосомы сои;
c) негенная последовательность имеет процентное содержание гуанина/цитозина в диапазоне от 25,62 до 43,76%; и,
d) негенная последовательность имеет длину приблизительно от 1 тпн до приблизительно 4,4 тпн.
II. Рекомбинантные производные идентифицированных оптимальных негенных геномных локусов сои
В соответствии с одним вариантом осуществления, после идентификации геномных локусов двудольного растения, такого как растение сои, в качестве наиболее подходящего местоположения для вставки донорных полинуклеотидных последовательностей, одна или более целевых нуклеиновых кислот могут быть вставлены в идентифицированный геномный локус. В одном варианте осуществления целевая нуклеиновая кислота включает экзогенные генные последовательности или другие нужные донорные полинуклеотидные последовательности. В другом варианте осуществления, после идентификации геномных локусов двудольного растения, такого как растение сои, в качестве наиболее подходящего местоположения для вставки донорных полинуклеотидных последовательностей, одна или более целевых нуклеиновых кислот оптимальных негенных геномных локусов сои необязательно могут быть удалены или вырезаны, с последующей интеграцией целевой ДНК в идентифицированный геномный локус. В одном варианте осуществления вставка целевой нуклеиновой кислоты в оптимальные негенные геномные локусы сои включает удаление, делецию или вырезание экзогенных генных последовательностей или других требуемых донорных полинуклеотидных последовательностей.
Настоящее описание также относится к способам и композициям для направленной интеграции в отобранный геномный локус сои с применением нуклеаз ZFN и конструкции донорного полинуклеотида. В способах вставки целевой последовательности нуклеиновой кислоты в оптимальные негенные геномные локусы сои, если не указано иное, используют стандартные методики в области молекулярной биологии, биохимии, структуры хроматина и анализа, клеточной культуры, рекомбинантной ДНК и связанных областях, как известно в уровне техники. Эти методики подробно описаны в литературе. См., например, Sambrook et al. MOLECULAR CLONING: A LABORATORY MANUAL, Second edition, Cold Spring Harbor Laboratory Press, 1989 и Third edition, 2001; Ausubel et al., CURRENT PROTOCOLS IN MOLECULAR BIOLOGY, John Wiley & Sons, New York, 1987, и периодические дополнения; серию METHODS IN ENZYMOLOGY, Academic Press, San Diego; Wolfe, CHROMATIN STRUCTURE AND FUNCTION, Third edition, Academic Press, San Diego, 1998; METHODS IN ENZYMOLOGY, Vol. 304, "Chromatin" (P. M. Wassarman and A. P. Wolffe, eds.), Academic Press, San Diego, 1999; и METHODS IN MOLECULAR BIOLOGY, Vol. 119, "Chromatin Protocols" (P. B. Becker, ed.) Humana Press, Totowa, 1999.
Способы вставки нуклеиновых кислот в геном сои
Любая из известных методик введения полинуклеотидных донорных последовательностей и последовательностей нуклеаз в виде конструкции ДНК в клетки-хозяева может применяться в соответствии с настоящим описанием. Они включают использование трансфекции с фосфатом кальция, полибреном, слияние протопластов, ПЭГ, электропорацию, ультразвуковые методы (например, сонопорацию), липосомы, микроинъекции, голую ДНК, плазмидные векторы, вирусные векторы, эписомные и интегративные, и любой из других известных методов введения клонированной геномной ДНК, кДНК, синтетической ДНК или другого чужеродного генетического материала в клетку-хозяина (см., например, Sambrook et al., выше). Необходимо только, чтобы конкретная используемая методика вставки нуклеиновых кислот позволяла успешно вводить в клетку-хозяина по меньшей мере один ген, способный экспрессировать выбранный белок.
Как отмечено выше, конструкции ДНК могут быть введены в геном требуемых видов растений с помощью множества стандартных методик. В качестве обзоров таких методик см., например, Weissbach & Weissbach Methods for Plant Molecular Biology (1988, Academic Press, N.Y.) Section VIII, стр. 421-463; и Grierson & Corey, Plant Molecular Biology (1988, 2d Ed.), Blackie, London, Гл. 7-9. Конструкция ДНК может быть введена непосредственно в геномную ДНК растительной клетки при использовании таких методик, как электропорация и микроинъекция протопластов растительных клеток, перемешивание с нитями карбида кремния (см., например, патенты США 5,302,523 и 5,464,765), или конструкции ДНК могут быть введены непосредственно в растительную ткань при использовании биолистических методов, таких как бомбардировка частицами с ДНК (см., например, Klein et al. (1987) Nature 327:70-73). В альтернативе конструкция ДНК может быть введена в растительную клетку посредством трансформации наночастицами (см., например, патентную публикацию США 20090104700, которая полностью включена в настоящую заявку посредством отсылки). В альтернативе конструкции ДНК могут быть объединены с подходящими T-ДНК бордерными/фланкирующими областями и введены в стандартный вектор для Agrobacterium tumefaciens. Опосредованные Agrobacterium tumefaciens методики трансформации, включая "разоружение" и использование бинарных векторов, хорошо описаны в научной литературе. См., например Horsch et al. (1984) Science 233:496-498, и Fraley et al. (1983) Proc. Nat'l. Acad. Sci. USA 80:4803.
Кроме того, перенос гена может быть выполнен при использовании других бактерий кроме агробактерии или вирусов, таких как Rhizobium sp. NGR234, Sinorhizoboium meliloti, Mesorhizobium loti, X вирус картофеля, вирус мозаики цветной капусты и вирус мозаики жилок маниока и/или вирус табачной мозаики, см., например, Chung et al. (2006) Trends Plant Sci. 11(1):1-4. Функции вирулентности Agrobacterium tumefaciens направляют вставку T-цепи, содержащей конструкцию и прилегающий маркер, в ДНК растительной клетки при заражении клетки бактериями с использованием бинарного T-ДНК вектора (Bevan (1984) Nuc. Acid Res. 12:8711-8721) или методики совместного культивирования (Horsch et al. (1985) Science 227:1229-1231). Как правило, систему агробактериальной трансформации используют в инженерии двудольных растений (Bevan et al. (1982) Ann. Rev. Genet. 16:357-384; Rogers et al. (1986) Methods Enzymol. 118:627-641). Система агробактериальной трансформации может также использоваться для трансформации, а также переноса ДНК в однодольные растения и растительные клетки. См. патент США 5,591,616; Hernalsteen et al. (1984) EMBO J. 3:3039-3041; Hooykass-Van Slogteren et al. (1984) Nature 311:763-764; Grimsley et al. (1987) Nature 325:1677-179; Boulton et al. (1989) Plant Mol. Biol. 12:31-40; и Gould et al. (1991) Plant Physiol. 95:426-434.
Альтернативные методы переноса генов и трансформации включают, без ограничения перечисленными, трансфомацию протопластов путем опосредованного хлоридом кальция, полиэтиленгликолем (ПЭГ) или электропорацией захвата голой ДНК (см. Paszkowski et al. (1984) EMBO J. 3:2717-2722, Potrykus et al. (1985) Molec. Gen. Genet. 199:169-177; Fromm et al. (1985) Proc. Nat. Acad. Sci. USA 82:5824-5828; и Shimamoto (1989) Nature 338:274-276) и электропорацию растительных тканей (D'Halluin et al. (1992) Plant Cell 4:1495-1505). Дополнительные способы трансформации растительных клеток включают микроинъекции, опосредованный карбидом кремния захват ДНК (Kaeppler et al. (1990) Plant Cell Reporter 9:415-418) и бомбардировку микрочастицами (см. Klein et al. (1988) Proc. Nat. Acad. Sci. USA 85:4305-4309; и Gordon-Kamm et al. (1990) Plant Cell 2:603-618).
В одном варианте осуществления целевая нуклеиновая кислота, вводимая в клетку-хозяина для направленной вставки в геном, включает гомологичные фланкирующие последовательности на одном или обоих концах направляемой целевой нуклеиновой кислоты. В таком варианте осуществления гомологичные фланкирующие последовательности содержат достаточные уровни идентичности последовательности по отношению к геномной последовательности двудольного растения, такой как геномная последовательность из сои, чтобы поддерживать гомологичную рекомбинацию между ней и геномной последовательностью, которой она гомологична. Приблизительно 25, 50, 100, 200, 500, 750, 1000, 1500 или 2000 нуклеотидов или более высокая идентичность последовательности, в пределах от 70% до 100%, между донорной и геномной последовательностью (или любое целочисленное значение в пределах 10-200 нуклеотидов или больше) поддерживает гомологичную рекомбинацию между ними.
В другом варианте осуществления направленная целевая нуклеиновая кислота не содержит гомологичных фланкирующих последовательностей, и направленная целевая нуклеиновая кислота обладает низкими или очень низкими уровнями идентичности последовательности с геномной последовательностью.
В других вариантах осуществления направленной рекомбинации и/или замены, и/или изменения последовательности в целевой области в клеточном хроматине, хромосомную последовательность изменяют посредством гомологичной рекомбинации с экзогенной "донорной" нуклеотидной последовательностью. Такую гомологичную рекомбинацию стимулирует присутствие двухцепочечного разрыва в клеточном хроматине, если присутствуют последовательности, гомологичные области разрыва. Двухцепочечные разрывы в клеточном хроматине могут также стимулировать клеточные механизмы негомологичного соединения концов. В любом из способов, описанных в настоящей заявке, первая нуклеотидная последовательность ("донорная последовательность") может содержать последовательности, которые являются гомологичными, но не идентичными, геномным последовательностям в целевой области, что стимулирует гомологичную рекомбинацию для вставки неидентичной последовательности в целевую область. Таким образом, в некоторых вариантах осуществления части донорной последовательности, которые гомологичны последовательностям в целевой области, демонстрируют от приблизительно 80, 85, 90, 95, 97,5 до 99% (или любое целое число между ними) идентичности последовательности с геномной последовательностью, которую заменяют. В других вариантах осуществления гомология между донорной и геномной последовательностью превышает 99%, например, если донорная и геномная последовательности отличаются только на 1 нуклеотид на протяжении 100 непрерывных пар оснований.
В некоторых случаях негомологичная часть донорной последовательности может содержать последовательности, которые не присутствуют в целевой области, в результате чего в целевую область вводятся новые последовательности. В таких случаях негомологичная последовательность обычно фланкирована последовательностями длиной 50-2000 пар оснований (или любое целочисленное значение между ними) или любым количеством пар оснований больше 2000, которые гомологичны или идентичны последовательностям в целевой области. В других вариантах осуществления донорная последовательность не гомологична целевой области и встраивается в геном посредством механизмов негомологичной рекомбинации.
В соответствии с одним вариантом осуществления цинк-пальцевая нуклеаза (ZFN) используется для введения двухцепочечного разрыва в геномный локус-мишень для облегчения вставки целевой нуклеиновой кислоты. Выбор сайта-мишени в выбранном геномном локусе для связывания доменом с цинковыми пальцами может быть выполнен, например, согласно способам, раскрытым в патенте США 6,453,242, описание которого включено в настоящую заявку, в данном патенте также раскрыты способы конструирования цинк-пальцевых белков (ZFP) для связывания с выбранной последовательностью. Специалистам в данной области будет ясно, что простой визуальный просмотр нуклеотидной последовательности может также использоваться для выбора сайта-мишени. Таким образом, в способах, описанных в настоящей заявке, могут использоваться любые средства для выбора сайта-мишени.
Что касается ZFP ДНК-связывающих доменов, сайты-мишени обычно состоят из множества примыкающих субсайтов-мишеней. Субсайт-мишень относится к последовательности, обычно триплету нуклеотидов или квадруплету нуклеотидов, которая может перекрываться на один нуклеотид с примыкающим квадруплетом, который связывает отдельный цинковый палец. См., например, WO 02/077227, описание которой включено в настоящую заявку. Сайт-мишень обычно имеет длину по меньшей мере 9 нуклеотидов и, соответственно, связывается цинк-пальцевым связывающим доменом, включающим по меньшей мере три цинковых пальца. Однако также возможно связывание, например, 4-пальцевого связывающего домена с сайтом-мишенью из 12 нуклеотидов, 5-пальцевого связывающего домена с сайтом-мишенью из 15 нуклеотидов или 6-пальцевого связывающего домена с сайтом-мишенью из 18 нуклеотидов. Как будет очевидно, связывание более крупных связывающих доменов (например, 7-, 8-, 9-пальцевых и больше) с более протяженными сайтами-мишенями также соответствует настоящему описанию.
В соответствии с одним вариантом осуществления сайт-мишень не должен быть кратным трем нуклеотидам. В случаях, в которых наблюдаются взаимодействия между цепями (см., например, патент США 6,453,242 и WO 02/077227), один или более отдельных цинковых пальцев многопальцевого связывающего домена может связываться с перекрывающимся квадруплетными субсайтами. В результате трехпальцевый белок может связываться с последовательностью из 10 нуклеотидов, в которой десятый нуклеотид является частью квадруплета, связываемого концевым пальцем, четырехпальцевый белок может связываться с последовательностью из 13 нуклеотидов, где тринадцатый нуклеотид является частью квадруплета, связываемого концевым пальцем, и т.д.
Длина и природа аминокислотных линкерных последовательностей между отдельными цинковыми пальцами в многопальцевом связывающем домене также влияет на связывание с последовательностью-мишенью. Например, наличие так называемого "неканонического линкера", "длинного линкера" или "структурного линкера" между смежными цинковыми пальцами в многопальцевом связывающем домене может позволять таким пальцам связывать субсайты, которые не примыкают друг к другу. Неограничивающие примеры таких линкеров описаны, например, в патенте США 6,479,626 и WO 01/53480. Соответственно, один или более субсайтов в сайте-мишени для цинк-пальцевого связывающего домена могут быть отделены друг от друга на 1, 2, 3, 4, 5 или более нуклеотидов. Одним из неограничивающих примеров может являться четырехпальцевый связывающий домен, который связывается с сайтом-мишенью из 13 нуклеотидов, включающим последовательно два смежных субсайта из 3 нуклеотидов, промежуточный нуклеотид и два смежных триплетных субсайта.
Хотя ДНК-связывающие полипептиды, идентифицированные из белков, которые существуют в природе, обычно связываются с дискретной нуклеотидной последовательностью или мотивом (например, консенсусной распознаваемой последовательностью), существуют и известны в уровне техники способы модификации многих таких ДНК-связывающих полипептидов, чтобы они распознавали другую нуклеотидную последовательность или мотив. ДНК-связывающие полипептиды включают, например, и без ограничения: цинк-пальцевые ДНК-связывающие домены; лейциновые молнии; UPA ДНК-связывающие домены; GAL4; TAL; LexA; Tet репрессор; LacR и рецептор стероидных гормонов.
В некоторых примерах ДНК-связывающий полипептид представляет собой цинковый палец. Могут быть созданы индивидуальные мотивы цинковых пальцев, которые направлены и специфично связываются с любым из целого ряда ДНК сайтов. Канонические Cys2His2 (а также неканонические Cys3His) цинк-пальцевые полипептиды связывают ДНК путем введения α-спирали в большую бороздку двойной спирали ДНК-мишени. Распознавание ДНК цинковым пальцем является модульным; каждый палец сначала входит в контакт с тремя последовательными парами оснований в мишени, при этом несколько ключевых остатков в полипептиде опосредуют распознавание. При включении множества цинк-пальцевых ДНК-связывающих доменов в направляющую эндонуклеазу, ДНК-связывающая специфичность направляющей эндонуклеазы может быть еще более повышена (и, следовательно, специфичность любых эффектов регуляции гена, придаваемая таким образом, может быть также повышена). См., например, Urnov et al. (2005) Nature 435:646-51. Таким образом, один или более цинк-пальцевых ДНК-связывающих полипептидов могут быть сконструированы и использованы таким образом, чтобы направляющая эндонуклеаза, вводимая в клетку-хозяина, взаимодействовала с последовательностью ДНК, которая является уникальной в геноме клетки-хозяина. Предпочтительно, цинк-пальцевый белок не встречается в природе, поскольку он сконструирован для связывания с выбранным сайтом-мишенью. См., например, Beerli et al. (2002) Nature Biotechnol. 20:135-141; Pabo et al. (2001) Ann. Rev. Biochem. 70:313-340; Isalan et al. (2001) Nature Biotechnol. 19:656-660; Segal et al. (2001) Curr. Opin. Biotechnol. 12:632-637; Choo et al. (2000) Curr. Opin. Struct. Biol. 10:411-416; патенты США 6,453,242; 6,534,261; 6,599,692; 6,503,717; 6,689,558; 7,030,215; 6,794,136; 7,067,317; 7,262,054; 7,070,934; 7,361,635; 7,253,273; и патентные публикации США 2005/0064474; 2007/0218528; 2005/0267061, полностью включенные в настоящую заявку посредством отсылки.
Сконструированный цинк-пальцевый связывающий домен может обладать новой специфичностью связывания по сравнению с природным цинк-пальцевым белком. Способы конструирования включают, без ограничения перечисленными, рациональное конструирование и различные типы отбора. Рациональное конструирование включает, например, использование баз данных, содержащих триплетные (или квадруплетные) нуклеотидные последовательности и индивидуальные аминокислотные последовательности цинковых пальцев, в которых каждая триплетная или квадруплетная нуклеотидная последовательность связана с одной или более аминокислотными последовательностями цинковых пальцев, которые связывают определенную триплетную или квадруплетную последовательность. См., например, находящиеся в совместном владении патенты США 6,453,242 и 6,534,261, полностью включенные в настоящую заявку посредством отсылки.
В альтернативе ДНК-связывающий домен может быть получен из нуклеазы. Например, известны распознаваемые последовательности хоуминг-эндонуклеаз и мегануклеаз, таких как I-SceI, I-CeuI, PI-PspI, PI-Sce, I-SceIV, I-CsmI, I-PanI, I-SceII, I-PpoI, I-SceIII, I-CreI, I-TevI, I-TevII и I-TevIII. См. также патент США 5,420,032; патент США 6,833,252; Belfort et al. (1997) Nucleic Acids Res. 25:3379–3388; Dujon et al. (1989) Gene 82:115–118; Perler et al. (1994) Nucleic Acids Res. 22, 1125–1127; Jasin (1996) Trends Genet. 12:224–228; Gimble et al. (1996) J. Mol. Biol. 263:163–180; Argast et al. (1998) J. Mol. Biol. 280:345–353 и каталог New England Biolabs. Кроме того, специфичность связывания ДНК хоуминг-эндонуклеаз и мегануклеаз может быть изменена с помощью генной инженерии для связывания неприродных сайтов-мишеней. См., например, Chevalier et al. (2002) Molec. Cell 10:895-905; Epinat et al. (2003) Nucleic Acids Res. 31:2952-2962; Ashworth et al. (2006) Nature 441:656-659; Paques et al. (2007) Current Gene Therapy 7:49-66; патентную публикацию США 20070117128.
В качестве другой альтернативы ДНК-связывающий домен может быть получен из белка лейциновой молнии. Лейциновые молнии представляют собой класс белков, которые участвуют в белок-белковых взаимодействиях многих эукариотических регуляторных белков, которые являются важными факторами транскрипции, связанными с экспрессией генов. Лейциновая молния относится к общему структурному мотиву, которым обладают такие факторы транскрипции из нескольких царств, включая животных, растений, дрожжей и т.д. Лейциновую молнию образуют два полипептида (гомодимер или гетеродимер), которые связываются с определенными последовательностями ДНК таким образом, что остатки лейцина равномерно распределяются по α-спирали, при этом остатки лейцина двух указанных полипептидов оказываются на одной стороне спирали. ДНК-связывающая специфичность лейциновых молний может быть использована в ДНК-связывающих доменах, раскрытых в настоящей заявке.
В некоторых вариантах осуществления ДНК-связывающий домен является сконструированным доменом из TAL-эффектора, полученного из фитопатогена Xanthomonas (см., Miller et al. (2011) Nature Biotechnology 29(2):143-8; Boch et al, (2009) Science 29 Oct 2009 (10.1126/science.117881) и Moscou and Bogdanove, (2009) Science 29 Oct 2009 (10.1126/science.1178817; а также патентные публикации США 20110239315, 20110145940 и 20110301073).
CRISPR (кластерные, разделенные регулярными интервалами, короткие палиндромные повторы)/Cas (CRISPR ассоциированная) нуклеазная система является недавно сконструированной нуклеазной системой, основанной на бактериальной системе, которая может применяться в геномной инженерии. Она основана на компоненте адаптивного иммунного ответа многих бактерий и археев. При проникновении вируса или плазмиды в бактерию, сегменты чужеродной ДНК превращаются в CRISPR РНК (crРНК) под воздействием 'иммунного' ответа. Такая crРНК затем связывается через область частичной комплементарности с другим типом РНК, называемой tracrРНК, которая направляет Cas9 нуклеазу к области в ДНК-мишени, называемой "протоспейсером" и гомологичной crРНК. Cas9 расщепляет ДНК с образованием тупых концов в DSB по сайтам, обозначенным 20-нуклеотидной направляющей последовательностью, содержащейся в транскрипте crРНК. Для сайт-специфического распознавания и расщепления ДНК Cas9 требуется crРНК и tracrРНК. Эту систему недавно сконструировали таким образом, чтобы crРНК и tracrРНК могли быть объединены в одну молекулу ("одиночная направляющая РНК"), при этом эквивалентная crРНК часть одиночной направляющей РНК может быть сконструирована так, чтобы направлять нуклеазу Cas9 для направленного воздействия на любую требуемую последовательность (см. Jinek et al (2012) Science 337, стр. 816-821, Jinek et al, (2013), eLife 2:e00471, и David Segal, (2013) eLife 2:e00563). Таким образом, система CRISPR/Cas может быть сконструирована таким способом, чтобы создавать двухцепочечный разрыв (DSB) в требуемой мишени в геноме, при этом на репарацию DSB может влиять применение ингибиторов репарации, чтобы вызвать увеличение репарации, склонной к ошибкам.
В некоторых вариантах осуществления белок Cas может быть "функциональным производным" природного белка Cas. "Функциональное производное" полипептида с нативной последовательностью является соединением, обладающим качественным биологическим свойством, общим с полипептидом, имеющим нативную последовательность. "Функциональные производные" включают, без ограничения перечисленными, фрагменты нативной последовательности и производные полипептида с нативной последовательностью, а также его фрагменты, при условии, что они обладают биологической активностью, общей с соответствующим полипептидом, имеющим нативную последовательность. Биологическая активность, рассматриваемая в настоящей заявке, является способностью функционального производного гидролизовать ДНК субстрат до фрагментов. Термин "производное" охватывает варианты аминокислотной последовательности полипептида, ковалентные модификации и их слияния. Подходящие производные полипептида Cas или его фрагмента включают, без ограничения перечисленными, мутанты, слияния, ковалентные модификации белка Cas или его фрагмента. Белок Cas, который включает белок Cas или его фрагмент, а также производные белка Cas или его фрагмента могут быть получены из клетки или синтезированы химически, или получены при объединении этих двух методик. Клетка может быть клеткой, которая естественно продуцирует белок Cas, или клеткой, которая естественно продуцирует белок Cas и генетически модифицирована с целью продукции эндогенного белка Cas с более высоким уровнем экспрессии или продукции белка Cas из вводимой извне нуклеиновой кислоты, которая кодирует Cas, который является таким же или отличается от эндогенного Cas. В некоторых случаях клетка в естественных условиях не продуцирует белок Cas и генетически модифицирована с целью продукции белка Cas. Белок Cas получают в клетках млекопитающих (и предположительно в растительных клетках) при коэкспрессии нуклеазы Cas с направляющей РНК. Для выполнения Cas-опосредованного расщепления генома могут использоваться две формы направляющих РНК, как раскрыто в Le Cong, F., et al., (2013) Science 339(6121):819-823.
В других вариантах осуществления ДНК-связывающий домен может быть связан с расщепляющим (нуклеазным) доменом. Например, хоуминг-эндонуклеазы могут быть модифицированы с изменением их ДНК-связывающей специфичности с одновременным сохранением их нуклеазной функции. Кроме того, цинк-пальцевые белки могут также быть слиты с расщепляющим доменом с получением цинк-пальцевой нуклеазы (ZFN). Часть расщепляющего домена слитых белков, раскрытых в настоящей заявке, может быть получена из любой эндонуклеазы или экзонуклеазы. Примеры эндонуклеаз, из которых может быть получен расщепляющий домен, включают, без ограничения перечисленными, некоторые эндонуклеазы рестрикции и хоуминг-эндонуклеазы. См., например, Catalogue 2002-2003, New England Biolabs, Beverly, MA; и Belfort et al. (1997) Nucleic Acids Res. 25:3379-3388. Известны дополнительные ферменты, которые расщепляют ДНК (например, нуклеаза S1; нуклеаза из бобов мунг; панкреатическая ДНКаза I; микрококковая нуклеаза; дрожжевая эндонуклеаза HO; см. также Linn et al., (eds.) Nucleases, Cold Spring Harbor Laboratory Press, 1993). Неограничивающие примеры хоуминг-эндонуклеаз и мегануклеаз включает I-SceI, I-CeuI, PI-PspI, PI-Sce, I-SceIV, I-CsmI, I-PanI, I-SceII, I-PpoI, I-SceIII, I-CreI, I-TevI, I-TevII и I-TevIII, которые известны. См. также патент США 5,420,032; патент США 6,833,252; Belfort et al. (1997) Nucleic Acids Res. 25:3379–3388; Dujon et al. (1989) Gene 82:115–118; Perler et al. (1994) Nucleic Acids Res. 22, 1125–1127; Jasin (1996) Trends Genet. 12:224–228; Gimble et al. (1996) J. Mol. Biol. 263:163–180; Argast et al. (1998) J. Mol. Biol. 280:345–353 и каталог New England Biolabs. Один или более таких ферментов (или их функциональных фрагментов) могут использоваться в качестве источника расщепляющих доменов и расщепляющих полудоменов.
Эндонуклеазы рестрикции (рестриктазы) присутствуют у многих видов и способны к сиквенс-специфическому связыванию с ДНК (на участке распознавания) и расщеплению ДНК на участке связывания или около него. Некоторые рестриктазы (например, типа IIS) расщепляют ДНК на участках, удаленных от участка распознавания, и имеют разделяемые связывающий и расщепляющий домены. Например, фермент типа IIS Fok I катализирует расщепление двухцепочечной ДНК на удалении 9 нуклеотидов от ее участка распознавания на одной цепи и 13 нуклеотидов от ее участка распознавания на другой цепи. См., например, патенты США 5,356,802; 5,436,150 и 5,487,994; а также Li et al. (1992) Proc. Natl. Acad. Sci. USA 89:4275-4279; Li et al. (1993) Proc. Natl. Acad. Sci. USA 90:2764-2768; Kim et al. (1994a) Proc. Natl. Acad. Sci. USA 91:883-887; Kim et al. (1994b) J. Biol. Chem. 269:31,978-31,982. Таким образом, в одном варианте осуществления слитые белки включают расщепляющий домен (или расщепляющий полудомен) по меньшей мере из одного рестрикционного фермента типа IIS и один или более цинк-пальцевых связывающих доменов, которые могут быть сконструированными или нет.
Примером рестрикционного фермента типа IIS, расщепляющий домен которого может быть отделен от связывающего домена, является Fok I. Этот специфический фермент активен в виде димера. Bitinaite et al. (1998) Proc. Natl. Acad. Sci. USA 95: 10,570-10,575. Соответственно, в рамках настоящего описания часть фермента Fok I, используемая в раскрытых слитых белках, считается расщепляющим полудоменом. Таким образом, для направленного двухцепочечного расщепления и/или направленной замены клеточных последовательностей с использованием слитых цинк-пальцевых-Fok I белков, два слитых белка, каждый из которых включает расщепляющий полудомен FokI, могут использоваться для воссоздания каталитически активного расщепляющего домена. В альтернативе может также использоваться одна полипептидная молекула, содержащая цинк-пальцевый связывающий домен и два расщепляющих полудомена Fok I. Параметры для направленного расщепления и направленного изменения последовательности с использованием слитых цинк-пальцевых-Fok I белков представлены в других частях настоящего описания.
Расщепляющий домен или расщепляющий полудомен могут являться любой частью белка, которая сохраняет расщепляющую активность или сохраняет способность к мультимеризации (например, димеризации) с образованием функционального расщепляющего домена. Примеры рестриктаз типа IIS описаны в международной публикации WO 2007/014275, полностью включенной в настоящую заявку посредством отсылки.
Для повышения специфичности расщепления расщепляющие домены также могут быть модифицированы. В некоторых вариантах осуществления используются варианты расщепляющего полудомена, которые минимизируют или предотвращают гомодимеризацию расщепляющих полудоменов. Неограничивающие примеры таких модифицированных расщепляющих полудоменов подробно описаны в WO 2007/014275, полностью включенной в настоящую заявку посредством отсылки. В некоторых вариантах осуществления расщепляющий домен включает сконструированный расщепляющий полудомен (также называемый димеризационно мутантным доменом), который минимизирует или предотвращает гомодимеризацию. Такие варианты осуществления известны специалистам в данной области и описаны, например, в патентных публикациях США 20050064474; 20060188987; 20070305346 и 20080131962, описания которых полностью включены в настоящую заявку посредством отсылки. Аминокислотные остатки в положениях 446, 447, 479, 483, 484, 486, 487, 490, 491, 496, 498, 499, 500, 531, 534, 537 и 538 FokI являются мишенями при воздействии на димеризацию расщепляющих полудоменов FokI.
Дополнительные сконструированные расщепляющие полудомены FokI, которые формируют облигатные гетеродимеры, также могут использоваться в ZFN, описанных в настоящей заявке. Примеры сконструированных расщепляющих полудоменов Fok I, которые формируют облигатные гетеродимеры, включают пару, в которой первый расщепляющий полудомен включает мутации по аминокислотным остаткам в положениях 490 и 538 Fok I, и второй расщепляющий полудомен включает мутации по аминокислотным остаткам 486 и 499. В одном варианте осуществления мутация в положении 490 приводит к замене Glu (E)→Lys (K); мутация в положении 538 приводит к замене Iso (I)→Lys (K); мутация в положении 486 приводит к замене Gln (Q)→Glu (E); и мутация в положении 499 приводит к замене Iso (I)→Lys (K). В частности, сконструированные расщепляющие полудомены, описанные в настоящей заявке, были получены в результате мутации в положениях 490 (E→K) и 538 (I→K) в одном расщепляющем полудомене, с получением сконструированного расщепляющего полудомена, обозначенного "E490K:I538K", и в результате мутации в положениях 486 (Q→E) и 499 (I→L) в другом расщепляющем полудомене, с получением сконструированного расщепляющего полудомена, обозначенного "Q486E:I499L". Сконструированные расщепляющие полудомены, описанные в настоящей заявке, являются мутантными облигатными гетеродимерами, в которых нарушение расщепления минимизировано или устранено. См., например, патентную публикацию США 2008/0131962, описание которой полностью включено посредством отсылки во всех отношениях. В некоторых вариантах осуществления сконструированный расщепляющий полудомен включает мутации в положениях 486, 499 и 496 (нумеруемых относительно FokI дикого типа), например мутации, которые приводят к замене остатка Gln (Q) дикого типа в положении 486 остатком Glu (E), остатка Iso (I) дикого типа в положении 499 остатком Leu (L) и остатка Asn (N) дикого типа в положении 496 остатком Asp (D) или Glu (E) (также обозначены как домены "ELD" и "ELE", соответственно). В других вариантах осуществления сконструированный расщепляющий полудомен включает мутации в положениях 490, 538 и 537 (нумеруемых относительно FokI дикого типа), например мутации, которые приводят к замене остатка Glu (E) дикого типа в положении 490 остатком Lys (K), остатка Iso (I) дикого типа в положении 538 остатком Lys (K) и остатка His (H) дикого типа в положении 537 остатком Lys (K) или Arg (R) (также обозначены как домены "KKK" и "KKR", соответственно). В других вариантах осуществления сконструированный расщепляющий полудомен включает мутации в положениях 490 и 537 (нумеруемых относительно FokI дикого типа), например мутации, которые приводят к замене остатка Glu (E) дикого типа в положении 490 остатком Lys (K) и остатка His (H) дикого типа в положении 537 остатком Lys (K) или Arg (R) (также обозначены как домены "KIK" и "KIR", соответственно). См. патентную публикацию США 20110201055. В других вариантах осуществления сконструированный расщепляющий полудомен включает мутации "Sharkey" и/или "Sharkey'" (см. Guo et al., (2010) J. Mol. Biol. 400(1):96-107).
Сконструированные расщепляющие полудомены, описанные в настоящей заявке, могут быть получены при использовании любого подходящего способа, например, с помощью сайт-направленного мутагенеза расщепляющих полудоменов дикого типа (Fok I), как описано в патентных публикациях США 20050064474; 20080131962 и 20110201055. В альтернативе нуклеазы могут собираться in vivo на сайте-мишени нуклеиновой кислоты при использовании так называемой технологии "сплит-энзим" (см. например, патентную публикацию США 20090068164). Компоненты таких сплит-энзимов могут экспрессироваться либо на отдельных экспрессионных конструкциях, либо могут быть связаны в одной открытой рамке считывания, где отдельные компоненты разделены, например, последовательностью саморасщепляющегося 2A пептида или IRES. Компоненты могут быть отдельными цинк-пальцевыми связывающими доменами или доменами мегануклеазы, связывающими нуклеиновую кислоту.
Нуклеазы могут быть подвергнуты скринингу на активность перед применением, например, в дрожжевой хромосомной системе, как описано в WO 2009/042163 и 20090068164. Экспрессионные конструкции нуклеазы могут быть с легкостью разработаны при использовании способов, известных в уровне техники. См., например, патентные публикации США 20030232410; 20050208489; 20050026157; 20050064474; 20060188987; 20060063231 и международную публикацию WO 07/014275. Экспрессия нуклеазы может находиться под контролем конститутивного промотора или индуцируемого промотора, например галактокиназного промотора, который активируется (дерепрессируется) в присутствии рафинозы и/или галактозы и репрессируется в присутствии глюкозы.
Расстояние между сайтами-мишенями относится к количеству нуклеотидов или пар нуклеотидов, расположенных между двумя сайтами-мишенями, при измерении от концов последовательностей, ближайших друг к другу. В некоторых вариантах осуществления, в которых расщепление зависит от связывания двух слитых молекул цинк-пальцевого домена/расщепляющего полудомена с отдельными сайтами-мишенями, два сайта-мишени могут находиться на разных цепях ДНК. В других вариантах осуществления оба сайта-мишени находятся на одной цепи ДНК. Для направленной интеграции в оптимальный геномный локус, одна или более ZFP являются сконструированными для связывания сайта-мишени в или около заданного сайта расщепления, при этом слитый белок, включающий сконструированный ДНК-связывающий домен и расщепляющий домен, экспрессируется в клетке. В результате связывания цинк-пальцевой части слитого белка с сайтом-мишенью, расщепляющий домен расщепляет ДНК, предпочтительно с образованием двухцепочечного разрыва, вблизи от сайта-мишени.
Наличие двухцепочечного разрыва в оптимальном геномном локусе способствует интеграции экзогенных последовательностей посредством гомологичной рекомбинации. Таким образом, в одном варианте осуществления полинуклеотид, включающий последовательность целевой нуклеиновой кислоты, встраиваемой в геномный локус-мишень, будет включать одну или более областей гомологии с геномным локусом-мишенью, способствующих гомологичной рекомбинации.
В дополнение к слитым молекулам, описанным в настоящей заявке, направленная замена выбранной геномной последовательности также включает введение донорной последовательности. Последовательность донорного полинуклеотида может быть введена в клетку до, одновременно или после экспрессии слитого белка(ов). В одном варианте осуществления донорный полинуклеотид обладает достаточной гомологией с оптимальным геномным локусом для поддержания гомологичной рекомбинации между ним и геномной последовательностью оптимального геномного локуса, которой он гомологичен. Гомологичную рекомбинацию будет поддерживать приблизительно 25, 50, 100, 200, 500, 750, 1000, 1500, 2000 нуклеотидов или более протяженная область гомологии последовательности между донорной и геномной последовательностью, или любое целочисленное значение от 10 до 2000 нуклеотидов или больше. В некоторых вариантах осуществления плечи гомологии имеют длину меньше 1000 пар оснований. В других вариантах осуществления плечи гомологии имеют длину меньше 750 пар оснований. В одном варианте осуществления донорные полинуклеотидные последовательности могут включать векторную молекулу, содержащую последовательности, которые не гомологичны целевой области в клеточном хроматине. Молекула донорного полинуклеотида может содержать несколько несплошных областей гомологии к клеточному хроматину. Например, для направленной вставки последовательностей, которые обычно не присутствуют в целевой области, указанные последовательности могут присутствовать в молекуле донорной нуклеиновой кислоты и могут быть фланкированы областями гомологии к последовательности в целевой области.
Донорный полинуклеотид может являться ДНК или РНК, одноцепочечной или двухцепочечной, и может быть введен в клетку в линейной или кольцевой форме. См., например, патентную публикацию США 20100047805, 20110281361, 20110207221 и заявку на патент США 13/889,162. В случае введения в линейной форме, концы донорной последовательности могут быть защищены (например, от экзонуклеазной деградации) с помощью методов, известных специалистам в данной области. Например, один или более дидезоксинуклеотидных остатков добавляют к 3’ концу линейной молекулы, и/или самокомплементарные олигонуклеотиды лигируют на одном или обоих концах. См., например, Chang et al. (1987) Proc. Natl. Acad. Sci. USA 84:4959-4963; Nehls et al. (1996) Science 272:886-889. Дополнительные методы защиты экзогенных полинуклеотидов от деградации включают, без ограничения перечисленными, присоединение концевой аминогруппы (аминогрупп) и использование модифицированных межнуклеотидных связей, таких как, например, фосфоротиоаты, фосфорамидаты и остатки O-метил-рибозы или дезоксирибозы.
В соответствии с одним вариантом осуществления предложен способ получения трансгенного двудольного растения, такого как растение сои, в котором целевая ДНК была вставлена в оптимальный негенный геномный локус сои. Способ включает следующие этапы:
a. выбор оптимального негенного локуса сои в качестве мишени для вставки целевой нуклеиновой кислоты;
b. введение сайт-специфической нуклеазы в клетку двудольного растения, такую как клетку растения сои, где сайт-специфическая нуклеаза расщепляет негенную последовательность;
c. введение целевой ДНК в клетку растения; и
d. отбор трансгенных растительных клеток, включающих целевую ДНК, направленную в указанную негенную последовательность.
В соответствии с одним вариантом осуществления предложен способ получения протопласта трансгенной клетки двудольного растения, такого как протопласт клетки сои, где целевая ДНК была вставлена в оптимальный негенный геномный локус сои. Способ включает следующие этапы:
a. выбор оптимального негенного локуса сои в качестве мишени для вставки целевой нуклеиновой кислоты;
b. введение сайт-специфической нуклеазы в протопласт клетки двудольного растения, такой как протопласт клетки сои, где сайт-специфическая нуклеаза расщепляет негенную последовательность;
c. введение целевой ДНК в протопласт клетки двудольного растения, такой как протопласт клетки сои; и
d. отбор протопласта трансгенной клетки двудольного растения, такого как протопласт клетки сои, включающего целевую ДНК, направленную в указанную негенную последовательность.
В одном варианте осуществления сайт-специфическая нуклеаза выбрана из группы, состоящей из цинк-пальцевой нуклеазы, нуклеазы CRISPR, нуклеазы TALEN или мегануклеазы, и, более конкретно, в одном варианте осуществления сайт-специфическая нуклеаза является цинк-пальцевой нуклеазой. В соответствии с одним вариантом осуществления целевая ДНК интегрируется в указанную негенную последовательность посредством способа интеграции, опосредованной направленной гомологией репарацией. В альтернативе, в некоторых вариантах осуществления целевая ДНК интегрируется в указанную негенную последовательность посредством способа интеграции, опосредованной негомологичным соединением концов. В дополнительных вариантах осуществления целевая ДНК интегрируется в указанную негенную последовательность посредством не описанного ранее способа интеграции. В одном варианте осуществления способ включает выбор оптимального негенного геномного локуса сои для направленной вставки целевой ДНК, который имеет 2, 3, 4, 5, 6, 7 или 8 из следующих характеристик:
a. негенная последовательность имеет длину по меньшей мере 1 тпн и не содержит более чем 1% метилирования ДНК в последовательности;
b. негенная последовательность демонстрирует частоту рекомбинации от 0,01574 до 83,52 сМ/мпн в геноме двудольного растения, таком как геном сои;
c. негенная последовательность демонстрирует уровень занятости нуклеосомами от 0 до 0,494 генома двудольного растения, такого как геном сои;
d. негенная последовательность обладает менее чем 40% идентичностью последовательности с любой другой последовательностью, содержащейся в геноме двудольного растения, таком как геном сои;
e. негенная последовательность имеет относительное значение местоположения от 0 до 0,99682 отношения геномного расстояния от центромеры хромосомны двудольного растения, такого как соя;
f. негенная последовательность имеет процентное содержание гуанина/цитозина в диапазоне от 14,4 до 45,9%;
g. негенная последовательность расположена проксимально к генной последовательности; и
h. 1 мпн область геномной последовательности двудольного растения, такой как геномная последовательность сои, включающая указанную негенную последовательность, включает одну или более дополнительных негенных последовательностей. В одном варианте осуществления оптимальный негенный локус сои выбран из локусов в кластере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 2, 3, 4, 5, 6, 7, 8, 9, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32.
Доставка
Донорные молекулы, раскрытые в настоящей заявке, интегрируются в геном клетки посредством направленных, независимых от гомологии или зависимых от гомологии способов. Для такой направленной интеграции геном расщепляют в нужном положении (или положениях) с использованием нуклеазы, например, слитых друг с другом ДНК-связывающего домена (например, цинк-пальцевый связывающий домен или TAL-эффекторный домен конструируют для связывания сайта-мишени в или вблизи заданного участка расщепления) и нуклеазного домена (например, расщепляющего домена или расщепляющего полудомена). В некоторых вариантах осуществления два слитых белка, каждый из которых включает ДНК-связывающий домен и расщепляющий полудомен, экспрессируются в клетке и связываются с сайтами-мишенями, которые расположены таким образом, что функциональный расщепляющий домен воссоздается, и ДНК расщепляется вблизи сайтов-мишеней. В одном варианте осуществления расщепление происходит между сайтами-мишенями двух ДНК-связывающих доменов. Один или оба ДНК-связывающих домена могут быть сконструированы. См. также патент США 7,888,121; патентную публикацию США 20050064474 и международные патентные публикации WO05/084190, WO05/014791 и WO 03/080809.
Нуклеазы, как описано в настоящей заявке, могут быть введены в виде полипептидов и/или полинуклеотидов. Например, два полинуклеотида, каждый из которых включает последовательности, кодирующие один из вышеуказанных полипептидов, могут быть введены в клетку, и когда полипептиды экспрессируются, и каждый из них связывается со своей целевой последовательностью, расщепление происходит в или вблизи последовательности-мишени. В альтернативе один полинуклеотид, включающий последовательности, кодирующие оба слитых полипептида, вводят в клетку. Полинуклеотиды могут представлять собой ДНК, РНК или их любые модифицированные формы или аналоги ДНК и/или РНК.
После введения двухцепочечного разрыва в целевой области трансген интегрируется в целевую область направленным способом, посредством независимых от гомологии методов (например, негомологичного соединения концов (NHEJ)), после линеаризации двухцепочечной донорной молекулы, как описано в настоящей заявке. Двухцепочечный донор предпочтительно линеаризуется in vivo нуклеазой, например, одной или более одинаковыми или различными нуклеазами, которые используются для введения двухцепочечного разрыва в геном. Синхронизированное расщепление хромосомы и донора в клетке может ограничивать деградацию донорной ДНК (по сравнению с линеаризацией донорной молекулы перед введением в клетку). Сайты-мишени нуклеазы, используемые для линеаризации донора, предпочтительно не прерывают последовательность(и) трансгена(ов).
Трансген может быть интегрирован в геном в направлении, предполагаемом при простом лигировании нуклеазных липких концов (обозначенном "прямая" или "AB" ориентация), или в другом направлении (обозначенном "обратная" или "BA" ориентация). В некоторых вариантах осуществления трансген интегрируется после точного лигирования липких концов хромосомы и донора. В других вариантах осуществления интеграция трансгена в BA или AB ориентации приводит к делеции нескольких нуклеотидов.
Посредством применения таких методик, как эти, могут быть стабильно трансформированы клетки практически любых видов. В некоторых вариантах осуществления трансформирующая ДНК интегрируется в геном клетки-хозяина. В случае многоклеточных видов трансгенные клетки могут быть регенерированы в трансгенный организм. Любая из этих методик может использоваться для получения трансгенного растения, например, включающего одну или более донорных полинуклеотидных последовательностей в геноме трансгенного растения.
Введение нуклеиновых кислот в растительную клетку в вариантах осуществления изобретения может быть выполнено любым способом, известным специалистам в данной области, включая, например, и без ограничения: трансформацию протопластов (См., например, патент США 5,508,184); опосредованный высыханием/ингибированием захват ДНК (см., например, Potrykus et al. (1985) Mol. Gen. Genet. 199:183-8); электропорацию (См., например, патент США 5,384,253); перемешивание с нитями карбида кремния (см., например, патенты США 5,302,523 и 5,464,765); опосредованную агробактерией трансформацию (см., например, патенты США 5,563,055, 5,591,616, 5,693,512, 5,824,877, 5,981,840 и 6,384,301); ускорение частиц, покрытых ДНК (см., например, патенты США 5,015,580, 5,550,318, 5,538,880, 6,160,208, 6,399,861 и 6,403,865) и наночастицы, наноносители и проникающие в клетку пептиды (WO201126644A2; WO2009046384A1; WO2008148223A1), в способах доставки ДНК, РНК, пептидов и/или белков или комбинаций нуклеиновых кислот и пептидов в растительные клетки.
Наиболее широко используемый метод введения вектора экспрессии в растения основан на природной системе трансформации агробактерий. A. tumefaciens и A. rhizogenes являются фитопатогенными почвенными бактериями, которые генетически трансформируют растительные клетки. Ti и Ri плазмиды A. tumefaciens и A. rhizogenes, соответственно, несут гены, ответственные за генетическую трансформацию растения. Ti (опухолеиндуцирующие)-плазмиды содержат крупный сегмент, известный как T-ДНК, который переносится в трансформированные растения. Другой сегмент Ti-плазмиды, vir-область, ответственен за перенос T-ДНК. Область T-ДНК ограничена левым и правым бордерами, каждый из которых состоит из концевых повторяющихся нуклеотидных последовательностей. В некоторых модифицированных бинарных векторах опухолеиндуцирующие гены были удалены, при этом функции vir-области используются для переноса чужеродной ДНК, ограниченной бордерными последовательностями T-ДНК. T-область также может содержать, например, селективный маркер для эффективного выделения трансгенных растений и клеток, а также сайт множественного клонирования для вставки последовательностей для переноса, таких как нуклеиновая кислота, кодирующая слитый белок согласно изобретению.
Таким образом, в некоторых вариантах осуществления вектор для трансформации растения получен из Ti-плазмиды A. tumefaciens (см., например, патенты США 4,536,475, 4,693,977, 4,886,937 и 5,501,967; и европейский патент EP 0 122 791) или Ri-плазмиды A. rhizogenes. Дополнительные векторы для трансформации растений включают, например, и без ограничения, векторы, описанные в Herrera-Estrella et al. (1983) Nature 303:209-13; Bevan et al. (1983), выше; Klee et al. (1985) Bio/Technol. 3:637-42; и в европейском патенте EP 0 120 516, а также полученные из любого из предыдущих. Другие бактерии, такие как Sinorhizobium, Rhizobium и Mesorhizobium, которые взаимодействуют с растениями в природе, могут быть модифицированы для опосредования переноса гена во многие различные растения. Такие фитоассоциированные симбиотические бактерии могут быть сделаны компетентными для переноса генов посредством приобретения разоруженной Ti-плазмиды и подходящего бинарного вектора.
Целевая нуклеиновая кислота
Донорные полинуклеотидные последовательности для направленной вставки в геномный локус двудольного растения, такого как растение сои, обычно имеют различную длину от приблизительно 10 до приблизительно 5000 нуклеотидов. Однако могут использоваться существенно более протяженные нуклеотиды, до 20000 нуклеотидов, включая последовательности длиной приблизительно 5, 6, 7, 8, 9, 10, 11 и 12 тпн. Кроме того, донорные последовательности могут включать векторную молекулу, содержащую последовательности, которые не гомологичны заменяемой области. В одном варианте осуществления целевая нуклеиновая кислота будет включать одну или более областей, которые обладают гомологией с направленными геномными локусами. Как правило, гомологичная область(и) последовательности целевой нуклеиновой кислоты будет обладать по меньшей мере 50% идентичностью последовательности с геномной последовательностью, с которой предполагается рекомбинация. В некоторых вариантах осуществления гомологичная область(и) целевой нуклеиновой кислоты обладает 60%, 70%, 80%, 90%, 95%, 98%, 99% или 99,9% идентичностью последовательности с последовательностями, расположенными в геномном локусе-мишени. Впрочем, может присутствовать любое значение между 1% и 100% идентичностью последовательности, в зависимости от длины целевой нуклеиновой кислоты.
Целевая нуклеиновая кислота может содержать несколько дискретных областей последовательности, обладающих относительно высокой идентичностью последовательности с клеточным хроматином. Например, для направленной вставки последовательностей, которые обычно не присутствуют в геномном локусе-мишени, в донорной молекуле нуклеиновой кислоты могут присутствовать уникальные последовательности, фланкированные областями последовательностей, которые обладают относительно высокой идентичностью последовательности с последовательностью, присутствующей в геномном локусе-мишени.
Целевая нуклеиновая кислота также может быть вставлена в направленный геномный локус, чтобы служить в качестве резервуара для последующего применения. Например, первая последовательность нуклеиновой кислоты, включающая последовательности, гомологичные негенной области генома двудольного растения, такого как растение сои, но содержащая целевую нуклеиновую кислоту (необязательно кодирующую ZFN под контролем индуцируемого промотора), может быть вставлена в направленный геномный локус. Затем вторую последовательность нуклеиновой кислоты вводят в клетку, чтобы вызвать вставку целевой ДНК в оптимальный негенный геномный локус двудольного растения, такого как растение сои. Либо первая последовательность нуклеиновой кислоты включает ZFN, специфичную к оптимальному негенному геномному локусу сои, а вторая последовательность нуклеиновой кислоты включают последовательность целевой ДНК, либо наоборот. В одном варианте осуществления ZFN расщепляет оптимальный негенный геномный локус сои и целевую нуклеиновую кислоту. Полученный в результате двухцепочечный разрыв в геноме может затем стать сайтом интеграции для целевой нуклеиновой кислоты, высвобождаемой из оптимального геномного локуса. В альтернативе экспрессия ZFN, уже расположенной в геноме, может быть индуцирована после введения целевой ДНК, чтобы вызвать образование в геноме двухцепочечного разрыва, который может затем стать сайтом интеграции для введенной целевой нуклеиновой кислоты. Таким образом, эффективность направленной интеграции целевой ДНК в любой целевой области может быть улучшена, поскольку способ не основан на одновременном введении нуклеиновых кислот, кодирующих ZFN, и целевой ДНК.
Целевая нуклеиновая кислота может быть также вставлена в оптимальный негенный геномный локус сои, чтобы служить в качестве сайта-мишени для последующих вставок. Например, в локус может быть вставлена целевая нуклеиновая кислота, состоящая из последовательностей ДНК, которые содержат сайты распознавания для дополнительных конструкций ZFN. Впоследствии дополнительные конструкции ZFN могут быть созданы и экспрессированы в клетках таким образом, чтобы исходная целевая нуклеиновая кислота была расщеплена и изменена посредством репарации или гомологичной рекомбинации. Таким образом, повторные интеграции целевой нуклеиновой кислоты могут происходить в оптимальном негенном геномном локусе двудольного растения, такого как растение сои.
Примеры экзогенных последовательностей, которые могут быть вставлены в оптимальный негенный геномный локус сои, включают, без ограничения перечисленными, любую кодирующую полипептид последовательность (например, кДНК), промотор, энхансер и другие регуляторные последовательности (например, последовательности интерферирующей РНК, кассеты экспрессии мшРНК, эпитопные метки, маркерные гены, сайты распознавания расщепляющих ферментов и различные типы экспрессионных конструкций. Такие последовательности могут быть с легкостью получены при использовании стандартных методик молекулярной биологии (клонирование, синтез и т.д.) и/или являются коммерчески доступными.
Для экспрессии ZFN, последовательности, кодирующие слитые белки, обычно субклонируют в вектор экспрессии, который содержит промотор, направляющий транскрипцию. Подходящие прокариотические и эукариотические промоторы известны в уровне техники и описаны, например, в Sambrook et al., Molecular Cloning, A Laboratory Manual (2nd ed. 1989; 3rd ed., 2001); Kriegler, Gene Transfer and Expression: A Laboratory Manual (1990); и Current Protocols in Molecular Biology (Ausubel et al., выше. Доступны бактериальные системы экспрессии для экспрессии ZFN, например, в E. coli, Bacillus sp. и Salmonella (Palva et al., Gene 22:229-235 (1983)). Наборы для таких систем экспрессии коммерчески доступны. Эукариотические системы экспрессии для клеток млекопитающих, дрожжей и клеток насекомых известны специалистам в данной области и также являются коммерчески доступными.
Конкретный вектор экспрессии, используемый для переноса генетического материала в клетку, выбирают с учетом предполагаемого применения слитых белков, например, экспрессии в растениях, животных, бактериях, грибах, одноклеточных и т.д. (см. векторы экспрессии, описанные ниже). Стандартные бактериальные и животные векторы экспрессии известны в уровне техники и подробно описаны, например, в патентной публикации США 20050064474A1 и международных патентных публикациях WO05/084190, WO05/014791 и WO03/080809.
Стандартные методы трансфекции могут использоваться для получения линий клеток бактерий, млекопитающих, дрожжей или насекомых, которые экспрессируют большие количества белка, который может быть затем очищен при использовании стандартных методик (см., например, ., Colley et al., J. Biol. Chem. 264:17619-17622 (1989); Guide to Protein Purification, in Methods in Enzymology, vol. 182 (Deutscher, ed., 1990)). Трансформацию эукариотических и прокариотических клеток выполняют согласно стандартным методикам (см., например, Morrison, J. Bact. 132:349-351 (1977); Clark-Curtiss & Curtiss, Methods in Enzymology 101:347-362 (Wu et al., eds., 1983)).
Раскрытые способы и композиции могут применяться для вставки донорных полинуклеотидных последовательностей в заданное положение, такое как один из оптимальных негенных геномных локусов сои. Это удобно, поскольку экспрессия введенного в геном сои трансгена крайне зависима от его участка интеграции. Таким образом, гены, кодирующие устойчивость к гербицидам, устойчивость к насекомым, питательные вещества, антибиотики или терапевтические молекулы, могут быть вставлены посредством направленной рекомбинации.
В одном варианте осуществления целевую нуклеиновую кислоту объединяют или "пакетируют" с кодирующими гены последовательностями, которые обеспечивают дополнительную устойчивость или невосприимчивость к глифосату или другому гербициду, и/или обеспечивают устойчивость к избранным насекомым или болезням, и/или повышают содержание питательных веществ, и/или улучшают агрономические характеристики, и/или белки или другие продукты, полезные при кормовом, пищевом, промышленном, фармацевтическом или другом применении. "Стэкинг" двух или более целевых последовательностей нуклеиновых кислот в геноме растения может быть выполнен, например, посредством обычной селекции растений с использованием двух или более объектов, трансформации растения конструкцией, которая содержит целевые последовательности, повторной трансформации трансгенного растения или добавления новых признаков с помощью направленной интеграции посредством гомологичной рекомбинации.
Такие нуклеотидные последовательности целевого донорного полинуклеотида включают, без ограничения перечисленными, примеры, представленные ниже:
1. Гены или кодирующая последовательность (например, иРНК), которые придают устойчивость к вредителям или болезням
(A) Гены устойчивости к болезням растений. Защита растений часто активируется при специфичном взаимодействии между продуктом гена устойчивости к болезни (R) в растении и соответствующим продуктом гена авирулентности (Avr) в патогене. Сорт растения может быть трансформирован клонированным геном устойчивости с получением сконструированных растений, которые устойчивы к определенным штаммам патогенов. Примеры таких генов включают ген Cf-9 томата для устойчивости к Cladosporium fulvum (Jones et al., 1994 Science 266:789), ген Pto томата, который кодирует протеинкиназу, для устойчивости к Pseudomonas syringae pv. tomato (Martin et al., 1993 Science 262:1432) и ген RSSP2 Arabidopsis для устойчивости к Pseudomonas syringae (Mindrinos et al., 1994 Cell 78:1089).
(B) Белок Bacillus thuringiensis, его производное или синтетический полипептид, моделируемый на их основе, такой как нуклеотидная последовательность гена δ-эндотоксина Bt (Geiser et al., 1986 Gene 48:109) и растительный инсектицидный ген (VIP) (см., например, Estruch et al. (1996) Proc. Natl. Acad. Sci. 93:5389-94). Кроме того, молекулы ДНК, кодирующие гены δ-эндотоксина, могут быть приобретены в Американской коллекции типовых культур (American Type Culture Collection, Rockville, Md.) под номерами ATCC 40098, 67136, 31995 и 31998.
(C) Лектин, такой как нуклеотидные последовательности нескольких генов маннозосвязывающих лектинов Clivia miniata (Van Damme et al., 1994 Plant Molec. Biol. 24:825).
(D) Витаминсвязывающий белок, такой как авидин и гомологи авидина, которые могут применяться в качестве ларвицидов против насекомых-вредителей. См. патент США 5,659,026.
(E) Ингибитор фермента, например, ингибитор протеазы или ингибитор амилазы. Примеры таких генов включают ингибитор цистеинпротеиназы риса (Abe et al., 1987 J. Biol. Chem. 262:16793), ингибитор протеиназы табака I (Huub et al., 1993 Plant Molec. Biol. 21:985) и ингибитор α-амилазы (Sumitani et al., 1993 Biosci. Biotech. Biochem. 57:1243).
(F) Специфичный для насекомых гормон или феромон, такой как экдистероид и ювенильный гормон, их вариант, миметик на их основе или их антагонист или агонист, например, бакуловирусная экспрессия клонированной эстеразы ювенильного гормона, инактиватора ювенильного гормона (Hammock et al., 1990 Nature 344:458).
(G) Специфичный для насекомых пептид или нейропептид, который при экспрессии нарушает физиологию обрабатываемого вредителя (J. Biol. Chem. 269:9). Примеры таких генов включают рецептор диуретического гормона насекомых (Regan, 1994), аллостатин, идентифицированный в Diploptera punctata (Pratt, 1989), и специфичные для насекомых паралитические нейротоксины (патент США 5,266,361).
(H) Специфичный для насекомых токсин, вырабатываемый в природе у змей, ос и т.д., такой как инсектотоксичный пептид скорпиона (Pang, 1992 Gene 116:165).
(I) Фермент, ответственный за избыточное накопление монотерпена, сесквитерпена, стероида, гидроксамовой кислоты, фенилпропаноидного производного или другой небелковой молекулы с инсектицидной активностью.
(J) Фермент, участвующий в модификации, в том числе посттрансляционной модификации, биологически активной молекулы; например, гликолитический фермент, протеолитический фермент, липолитический фермент, нуклеаза, циклаза, трансаминаза, эстераза, гидролаза, фосфатаза, киназа, фосфорилаза, полимераза, эластаза, хитиназа и глюканаза, природные или синтетические. Примеры таких генов включают ген callas (опубликованная заявка PCT WO93/02197), кодирующие хитиназу последовательности (которые могут быть получены, например, из ATCC под номерами 3999637 и 67152), хитиназу табачного бражника (Kramer et al., 1993 Insect Molec. Biol. 23:691) и ген полиубиквитина ubi4-2 петрушки (Kawalleck et al., 1993 Plant Molec. Biol. 21:673).
(K) Молекула, которая стимулирует передачу сигналов. Примеры таких молекул включают нуклеотидные последовательности кДНК клонов кальмодулина бобов мунг (Botella et al., 1994 Plant Molec. Biol. 24:757) и нуклеотидную последовательность кДНК клона кальмодулина кукурузы (Griess et al., 1994 Plant Physiol. 104:1467).
(L) Пептид с гидрофобным моментом. См. патенты США 5,659,026 и 5,607,914; в последнем описаны синтетические противомикробные пептиды, которые придают устойчивость к болезням.
(M) Мембранная пермеаза, формирователь каналов или блокатор каналов, такой как аналог цекропин-β литического пептида (Jaynes et al., 1993 Plant Sci. 89:43), который делает трансгенные растения табака устойчивыми к Pseudomonas solanacearum.
(N) Вирусно-инвазивный белок или сложный токсин, получаемый из него. Например, накопление вирусных белков оболочки в трансформированных растительных клетках придает устойчивость к вирусной инфекции и/или развитию болезни, вызываемой вирусом, из которого получен ген белка оболочки, а также родственными вирусами. Опосредованную белком оболочки устойчивость придавали трансформированным растениям против вируса мозаики люцерны, вируса мозаики огурца, вируса полосатости табака, вируса X картофеля, вируса Y картофеля, вируса гравировки табака, вируса погремковости табака и вируса табачной мозаики. См., например, Beachy et al. (1990) Ann. Rev. Phytopathol. 28:451.
(O) Специфичное для насекомых антитело или полученный из него иммунотоксин. Так, например, антитело, направленное против важной метаболической функции в кишечнике насекомого, будет инактивировать соответствующий фермент, уничтожая насекомое. Например, в Taylor et al. (1994) Abstract #497, Seventh Int'l Symposium on Molecular Plant-Microbe Interactions, показана ферментативная инактивация в трансгенном табаке в результате продукции одноцепочечных фрагментов антитела.
(P) Вирусспецифичное антитело. См., например, Tavladoraki et al. (1993) Nature 266:469, где показано, что трансгенные растения, экспрессирующие рекомбинантные гены антител, защищены от вирусной атаки.
(Q) Ингибирующий развитие белок, вырабатываемый в природе патогеном или паразитом. Так, например, грибковые эндо-α-1,4-D полигалактуроназы способствуют грибковой колонизации и высвобождению питательных веществ из растения в результате солюбилизации стенки растительных клеток гомо-α-1,4-D-галактуроназой (Lamb et al., 1992) Bio/Technology 10:1436. Клонирование и исследование гена, который кодирует эндополигалактуроназа-ингибирующий белок фасоли, описаны в Toubart et al. (1992 Plant J. 2:367).
(R) Ингибирующий развитие белок, вырабатываемый в природе растением, такой как рибосома-инактивирующий ген ячменя, который обеспечивает повышенную устойчивость к грибковой болезни (Longemann et al., 1992). Bio/Technology 10:3305.
(S) РНК интерференция, в которой молекула РНК используется для ингибирования экспрессии гена-мишени. Молекула РНК в одном примере является частично или полностью двухцепочечной, что вызывает реакцию сайленсинга, который приводит к расщеплению дцРНК на малые интерферирующие РНК, которые затем включаются в направляющий комплекс, который разрушает гомологичные мРНК. См., например, Fire et al., патент США 6,506,559; Graham et al. 6,573,099.
2. Гены, которые придают устойчивость к гербициду
(A) Гены, кодирующие устойчивость или невосприимчивость к гербициду, который ингибирует точку роста или меристему, такому как имидазалиноновый, сульфонанилидный или сульфонилмочевинный гербицид. Примеры генов в этой категории кодируют мутантную ацетолактатсинтазу (ALS) (Lee et al., 1988 EMBOJ. 7:1241), которая также известна как фермент синтаза ацетогидроксикислот (AHAS) (Miki et al., 1990 Theor. Appl. Genet. 80:449).
(B) Один или более дополнительных генов, кодирующих устойчивость или невосприимчивость к глифосату, придаваемую мутантными генами EPSP синтазы и aroA, или в результате метаболической инактивации такими генами, как DGT-28, 2mEPSPS, GAT (глифосат-ацетилтрансфераза) или GOX (глифосатоксидаза), а также к другим фосфоновым соединениям, таким как глуфосинат (гены pat, bar и dsm-2), и арилоксифеноксипропионовым кислотам и циклогександионам (гены, кодирующие ингибитор ацетил-КоА-карбоксилазы). См., например, патент США 4,940,835, в котором раскрыта нуклеотидная последовательность формы EPSP, которая может придавать устойчивость к глифосату. Молекула ДНК, кодирующая мутантный ген aroA, может быть получена под номером ATCC 39256, и нуклеотидная последовательность мутантного гена раскрыта в патенте США 4,769,061. В европейской заявке на патент 0 333 033 и патенте США 4,975,374 раскрыты нуклеотидные последовательности генов глутаминсинтетазы, которые придают устойчивость к гербицидам, таким как L-фосфинотрицин. Нуклеотидная последовательность гена фосфинотрицинацетил-трансферазы приведена в европейской заявке 0 242 246. В De Greef et al. (1989) Bio/Technology 7:61 описано получение трансгенных растений, которые экспрессируют химерные bar гены, кодирующие активность фосфинотрицинацетилтрансферазы. Примерами генов, придающих устойчивость к арилоксифеноксипропионовым кислотам и циклогександионам, таким как сетоксидим и галоксифоп, являются гены Accl-S1, Accl-S2 и Accl-S3, описанные в Marshall et al. (1992) Theor. Appl. Genet. 83:435.
(C) Гены, кодирующие устойчивость или невосприимчивость к гербициду, который ингибирует фотосинтез, такому как триазин (гены psbA и gs+) и бензонитрил (ген нитрилазы). В Przibilla et al. (1991) Plant Cell 3:169 описано применение плазмид, кодирующих мутантные гены psbA, для трансформации Chlamydomonas. Нуклеотидные последовательности генов нитрилазы раскрыты в патенте США 4,810,648, и молекулы ДНК, содержащие эти гены, доступны под номерами ATCC 53435, 67441 и 67442. Клонирование и экспрессия ДНК, кодирующей глутатион-S-трансферазу, описаны в Hayes et al. (1992) Biochem. J. 285:173.
(D) Гены, кодирующие устойчивость или невосприимчивость к гербициду, который связывается с гидроксифенилпируват-диоксигеназами (HPPD), ферментами, которые катализируют реакцию, в которой пара-гидроксифенилпируват (HPP) превращается в гомогентизат. Это включает гербициды, такие как изоксазолы (EP418175, EP470856, EP487352, EP527036, EP560482, EP682659, патент США 5,424,276), в частности изоксафлутол, который является селективным гербицидом для сои, дикетонитрилы (EP496630, EP496631), в частности 2-циано-3-циклопропил-1-(2-SO2CH3-4-CF3-фенил)пропан-1,3-дион и 2-циано-3-циклопропил-1-(2-SO2CH3-4-2,3Cl2фенил)пропан-1,3-дион, трикетоны (EP625505, EP625508, патент США 5,506,195), в частности сулкотрион, и пиразолинаты. Ген, который производит избыток HPPD в растениях, может придавать невосприимчивость или устойчивость к таким гербицидам, включая, например, гены, описанные в патентах США 6,268,549 и 6,245,968 и публикации заявки на патент США 20030066102.
(E) Гены, кодирующие устойчивость или невосприимчивость к феноксиауксиновым гербицидам, таким как 2,4-дихлорфеноксиуксусная кислота (2,4-D), и которые могут также придавать устойчивость или невосприимчивость к арилоксифеноксипропионатным (AOPP) гербицидам. Примеры таких генов включают ген фермента α-кетоглутарат-зависимой диоксигеназы (aad-1), описанный в патенте США 7,838,733.
(F) Гены, кодирующие устойчивость или невосприимчивость к феноксиауксиновым гербицидам, таким как 2,4- дихлорфеноксиуксусная кислота (2,4-D), и которые могут также придавать устойчивость или невосприимчивость к пиридилоксиауксиновым гербицидам, таким как флуроксипир или триклопир. Примеры таких генов включают ген фермента α-кетоглутарат-зависимой диоксигеназы (aad-12), описанный в WO 2007/053482 A2.
(G) Гены, кодирующие устойчивость или невосприимчивость к дикамбе (см., например, патентную публикацию США 20030135879).
(H) Гены, обеспечивающие устойчивость или невосприимчивость к гербицидам, которые ингибируют протопорфириногеноксидазу (PPO) (см. патент США 5,767,373).
(I) Гены, обеспечивающие устойчивость или невосприимчивость к триазиновым гербицидам (таким как атразин) и гербицидам производным мочевины (такие как диурон), которые связываются с кор-белками реакционных центров фотосистемы II (PS II) (см. Brussian et al., (1989) EMBO J. 1989, 8(4): 1237-1245).
3. Гены, которые придают или способствуют приобретению ценного признака
(A) Модифицированный метаболизм жирных кислот, например, при трансформации сои или Brassica антисмысловым геном стеароил-АПБ-десатуразы с увеличением содержания стеариновой кислоты в растении (Knultzon et al., 1992 Proc. Nat. Acad. Sci. USA 89:2624).
(B) Уменьшенное содержание фитата
(1) Введение кодирующего фитазу гена, такого как ген фитазы Aspergillus niger (Van Hartingsveldt et al., 1993 Gene 127:87), усиливает расщепление фитата, добавляя больше свободного фосфата в трансформированное растение.
2) Может быть введен ген, который уменьшает содержание фитата. В двудольных растениях это можно осуществить, например, путем клонирования и последующего повторного введения ДНК, ассоциированной с одним аллелем, который может быть ответственным за мутанты сои, характеризующиеся низкими уровнями фитиновой кислоты (Raboy et al., 1990 Maydica 35:383).
(C) Модифицированный углеводный состав, получаемый, например, при трансформации растений геном, кодирующим фермент, который изменяет структуру разветвления крахмала. Примеры таких ферментов включают ген фруктозилтрансферазы Streptococcus mucus (Shiroza et al., 1988, J. Bacteol. 170:810), ген левансахаразы Bacuillus subtilis (Steinmetz et al., 1985 Mol. Gen. Genel. 200:220), α-амилазу Bacillus licheniformis (Pen et al., 1992 Bio/Technology 10:292), гены инвертазы томата (Elliot et al., 1993), ген амилазы ячменя (Sogaard et al., 1993 J. Biol. Chem. 268:22480) и крахмал-разветвляющий фермент II эндосперма сои (Fisher et al., 1993 Plant Physiol. 102:10450).
III. Рекомбинантные конструкции
Как раскрыто в настоящей заявке, в настоящем описании предложены рекомбинантные геномные последовательности, включающие оптимальную негенную геномную последовательность сои длиной по меньшей мере 1 тпн и целевую ДНК, где вставленная целевая ДНК вставлена в указанную негенную последовательность. В одном варианте осуществления целевая ДНК является аналитическим доменом, геном или кодирующей последовательностью (например, иРНК), которые придают устойчивость к вредителям или болезни, генами, которые придают устойчивость к гербициду, или генами, которые придают или способствуют приобретению ценного признака, при этом оптимальная негенная геномная последовательность сои включает 1, 2, 3, 4, 5, 6, 7 или 8 из следующих характеристик:
a. негенная последовательность имеет длину от приблизительно 1 тпн до приблизительно 5,7 тпн и не содержит метилированный полинуклеотид;
b. негенная последовательность демонстрирует частоту рекомбинации от 0,01574 до 83,52 сМ/мпн в геноме двудольного растения, такого как растение сои;
c. негенная последовательность демонстрирует уровень занятости нуклеосомами от 0 до 0,494 генома двудольного растения, такого как геном сои;
d. негенная последовательность обладает менее чем 40% идентичностью последовательности с любой другой последовательностью, содержащейся в геноме двудольного растения, таком как геном сои;
e. негенная последовательность имеет относительное значение местоположения от 0 до 0,99682 отношения геномного расстояния от центромеры хромосомы двудольного растения, такой как центр хромосомы сои;
f. негенная последовательность имеет процентное содержание гуанина/цитозина в диапазоне от 14,4 до 45,9%;
g. негенная последовательность расположена проксимально к генной последовательности, включающей известную или предсказанную кодирующую последовательность двудольного растения, такую как кодирующая последовательность сои, в пределах 40 тпн примыкающей геномной ДНК, включающей нативную негенную последовательность; и
h. негенная последовательность расположена в 1 мпн области геномной последовательности двудольного растения, такой как геномная последовательность, которая включает по меньшей мере вторую негенную последовательность.
В одном варианте осуществления оптимальная негенная геномная последовательность сои дополнительно отличается наличием генной области, включающей 1-18 известных или предсказанных кодирующих последовательностей сои в пределах 40 тпн примыкающей геномной ДНК, включающей нативную негенную последовательность. В одном варианте осуществления оптимальный негенный локус сои выбран из локусов в кластере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 2, 3, 4, 5, 6, 7, 8, 9, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31 или 32.
IV. Трансгенные растения
Трансгенные растения, включающие рекомбинантные оптимальные негенные локусы сои, также предложены в соответствии с одним вариантом осуществления настоящего описания. Такие трансгенные растения могут быть получены с применением методик, известных специалистам в данной области.
Трансформированная клетка двудольного растения, каллус, ткань или растение (то есть клетка, каллус, ткань или растение сои) могут быть идентифицированы и выделены посредством отбора или скрининга сконструированного растительного материала на наличие признаков, кодируемых маркерными генами, присутствующими в трансформирующей ДНК. Например, отбор может быть выполнен посредством выращивания сконструированного растительного материала на среде, содержащей ингибирующее количество антибиотика или гербицида, к которым придает устойчивость трансформирующая генная конструкция. Кроме того, трансформированные клетки могут быть также идентифицированы с помощью скрининга на активность любых видимых маркерных генов (например, желтого флуоресцентного белка, зеленого флуоресцентного белка, красного флуоресцентного белка, бета-глюкуронидазы, люциферазы, генов B или C1), которые могут присутствовать в рекомбинантных конструкциях нуклеиновых кислот. Такие методики отбора и скрининга известны специалистам в данной области.
Физические и биохимические методы также могут использоваться для идентификации растения или растительных клеток-трансформантов, содержащих вставленные генные конструкции. Такие методы включают, без ограничения перечисленными: 1) Саузерн-анализ или ПЦР-амплификацию для обнаружения и определения структуры вставки рекомбинантной ДНК; 2) Нозерн-блот, защиту от S1 РНКазы, удлинение праймеров или ПЦР-амплификацию с обратной транскриптазой для обнаружения и исследования РНК-транскриптов генных конструкций; 3) ферментативные анализы для обнаружения активности фермента или рибозима, где такие продукты генов кодируются генной конструкцией; 4) белковый гель-электрофорез, методики Вестерн-блоттинга, иммунопреципитация или иммуноферментные анализы (ELISA), где продуктами генных конструкций являются белки. Дополнительные методики, такие как in situ гибридизация, ферментное окрашивание и иммунное окрашивание, также могут использоваться для обнаружения присутствия или экспрессии рекомбинантной конструкции в определенных органах и тканях растений. Способы проведения всех таких анализов известны специалистам.
Результаты манипуляции генами с применением способов, раскрытых в настоящем описании, можно наблюдать, например, с помощью Нозерн-блотов РНК (например, мРНК), выделенной из целевых тканей. Как правило, если мРНК присутствует, или количество мРНК возросло, можно предположить, что соответствующий трансген экспрессируется. Могут использоваться другие методы измерения активности гена и/или кодируемого полипептида. Могут использоваться различные типы ферментных анализов в зависимости от используемого субстрата и метода обнаружения увеличения или уменьшения продукта реакции или побочного продукта. Кроме того, уровни экспрессируемого полипептида могут быть измерены иммунохимически, то есть с помощью ELISA, RIA, EIA и других анализов на основе антител, известных специалистам в данной области, таких как электрофоретические анализы детектирования (с окрашиванием или вестерн-блоттингом). В качестве одного из неограничивающих примеров, детектирование AAD-12 (арилоксиалканоатдиоксигеназы; см. WO 2011/066360) и PAT (фосфинотрицин-N-ацетилтрансферазы (PAT)) белков с использованием анализа ELISA описано в патентной публикации США 20090093366, которая полностью включена в настоящую заявку посредством отсылки. Трансген может выборочно экспрессироваться в некоторых тканях растения или на некоторых стадиях развития, или трансген может экспрессироваться практически во всех тканях растения, по существу на протяжении всего его жизненного цикла. Впрочем, также может применяться любой комбинаторный режим экспрессии.
Специалисту в данной области известно, что после стабильного включения экзогенной полинуклеотидной донорной последовательности в трансгенные растения и подтверждения ее функциональности она может быть введена в другие растения при половом скрещивании. Может использоваться любая стандартная методика скрещивания в зависимости от скрещиваемых видов.
Настоящее описание также охватывает семена трансгенных растений, описанных выше, где семя содержит трансген или генную конструкцию. Настоящее описание также охватывает потомство, клоны, клеточные линии или клетки трансгенных растений, описанных выше, где потомство, клон, клеточная линия или клетки содержат трансген или генную конструкцию, вставленные в оптимальные геномные локусы.
Трансформированные растительные клетки, которые получены с помощью любой из вышеуказанных методик трансформации, можно культивировать с регенерацией целого растения, которое обладает трансформированным генотипом и, таким образом, требуемым фенотипом. Такие методики регенерации основаны на манипуляции некоторыми фитогормонами в среде для выращивания культур тканей, которые обычно основаны на применении биоцидного и/или гербицидного маркера, который был введен вместе с нужными нуклеотидными последовательностями. Регенерация растения из культивируемых протопластов описана в Evans, et al., "Protoplasts Isolation and Culture", в Handbook of Plant Cell Culture, стр. 124-176, Macmillian Publishing Company, New York, 1983; и Binding, Regeneration of Plants, Plant Protoplasts, стр. 21-73, CRC Press, Boca Raton, 1985. Регенерация также может быть получена из растительного каллуса, эксплантатов, органов, пыльцы, зародышей или их частей. Такие методики регенерации описаны в общем виде в Klee et al. (1987) Ann. Rev. of Plant Phys. 38:467-486.
Трансгенное растение или растительный материал, включающие нуклеотидные последовательности, кодирующие полипептид, в некоторых вариантах осуществления могут демонстрировать одно или более следующих свойств: экспрессия полипептида в клетке растения; экспрессия части полипептида в пластиде клетки растения; импорт полипептида из цитозоля клетки растения в пластиду клетки; пластида-специфическая экспрессия полипептида в клетке растения; и/или локализация полипептида в клетке растения. Такое растение может дополнительно обладать одним или более требуемыми признаками помимо экспрессии кодируемого полипептида. Такие признаки могут включать, например: устойчивость к насекомым, другим вредителям и болезнетворным агентам; невосприимчивость к гербицидам; повышенную устойчивость, урожайность или продолжительность хранения; устойчивость к воздействию окружающей среды; продукцию фармацевтических продуктов; продукцию промышленных продуктов; и повышенную пищевую ценность.
В соответствии с одним вариантом осуществления предложен протопласт трансгенного двудольного растения (то есть протопласт сои), включающий рекомбинантный оптимальный негенный локус сои. Более конкретно, предложен протопласт двудольного растения, такой как протопласт сои, включающий целевую ДНК, вставленную в оптимальные негенные геномные локусы сои протопласта двудольного растения (то есть протопласта сои), где указанные негенные геномные локусы сои имеют длину от приблизительно 1 тпн до приблизительно 5,7 тпн и не содержат метилированных нуклеотидов. В одном варианте осуществления протопласт трансгенного двудольного растения (то есть протопласт трансгенной сои) включает целевую ДНК, вставленную в оптимальный негенный геномный локус сои, где целевая ДНК включает аналитический домен и/или открытую рамку считывания. В одном варианте осуществления вставленная целевая ДНК кодирует пептид, а в другом варианте осуществления целевая ДНК включает по меньшей мере одну кассету экспрессии гена, включающую трансген.
В соответствии с одним вариантом осуществления предложены трансгенное двудольное растение, часть двудольного растения или растительная клетка двудольного растения (то есть трансгенное растение сои, часть растения сои или клетка растения сои), включающие рекомбинантный оптимальный негенный локус сои. Более конкретно, предложены двудольное растение, часть двудольного растения или клетка двудольного растения (то есть растение сои, часть растения сои или клетка растения сои), включающие целевую ДНК, вставленную в оптимальные негенные геномные локусы сои двудольного растения, части двудольного растения или клетки двудольного растения (то есть растения сои, части растения сои или клетки растения сои), где указанные негенные геномные локусы сои имеют длину от приблизительно 1 тпн до приблизительно 5,7 тпн и не содержат метилированных нуклеотидов. В одном варианте осуществления трансгенное двудольное растение, часть двудольного растения или клетка двудольного растения (то есть трансгенное растение сои, часть растения сои или клетка растения сои) включают целевую ДНК, вставленную в оптимальный негенный геномный локус сои, где целевая ДНК включает аналитический домен и/или открытую рамку считывания. В одном варианте осуществления вставленная целевая ДНК кодирует пептид, а в другом варианте осуществления целевая ДНК включает по меньшей мере одну кассету экспрессии гена, включающую трансген.
В соответствии с вариантом осуществления 1 предложена рекомбинантная последовательность, где негенная геномная последовательность сои длиной по меньшей мере 1 тпн, где указанная негенная последовательность является гипометилированной, может служить в качестве мишени, расположена проксимально к генной области в геноме сои и демонстрирует подтверждение рекомбинации, дополнительно включает целевую ДНК, вставленную в указанную негенную последовательность. В соответствии с вариантом осуществления 2 рекомбинантная последовательности согласно варианту осуществления 1 имеет следующие характеристики:
a. уровень метилирования указанной негенной последовательности составляет 1% или меньше;
b. указанная негенная последовательность обладает менее чем 40% идентичностью последовательности с любой другой последовательностью, содержащейся в геноме сои;
c. указанная негенная последовательность расположена в пределах 40 тпн области известной или предсказанной экспрессируемой кодирующей последовательности сои; и
d. указанная негенная последовательность демонстрирует частоту рекомбинации в геноме сои больше 0,01574 сМ/мпн. В соответствии с вариантом осуществления 3 предложена рекомбинантная последовательность согласно варианту осуществления 1 или 2, где указанная негенная последовательность включает максимальную длину 5,73 тпн. В соответствии с вариантом осуществления 4 предложена рекомбинантная последовательность согласно любому из вариантов осуществления 1-3, где указанная негенная последовательность включает 1% или меньше метилирования нуклеотидов. В соответствии с вариантом осуществления 5 предложена рекомбинантная последовательность согласно любому из вариантов осуществления 1-4, где указанная негенная последовательность имеет длину от 1 тпн до 5,73 тпн и не содержит метилированных остатков цитозина. В соответствии с вариантом осуществления 6 предложена рекомбинантная последовательность согласно любому из вариантов осуществления 1-5, где указанная негенная последовательность не выравнивается с более чем 40% идентичностью последовательности ни с одной другой последовательностью в геноме сои.
В соответствии с вариантом осуществления 7 предложена рекомбинантная последовательность согласно любому из пп. 1-6, где указанная негенная последовательность представляет подтверждение рекомбинации с частотой рекомбинации больше 0,01574 сМ/мпн. В соответствии с вариантом осуществления 8 предложена рекомбинантная последовательность согласно любому из пп. 1-7, где 40 тпн область нативного генома сои, включающая указанную негенную последовательность, также включает по меньшей мере одну известную или предсказанную кодирующую последовательность сои или последовательность, включающую последовательность 2 тпн до и/или 1 тпн после известного гена сои. В соответствии с вариантом осуществления 9 предложена рекомбинантная последовательность согласно любому из пп. 1-8, где указанная известная или предсказанная кодирующая последовательность сои экспрессирует белок сои. В соответствии с вариантом осуществления 10 предложена рекомбинантная последовательность согласно любому из пп. 1-9, где указанная негенная последовательность не содержит метилированный полинуклеотид. В соответствии с вариантом осуществления 11 предложена рекомбинантная последовательность согласно любому из пп. 1-10, где один конец указанной негенной последовательности расположен в пределах 40 тпн от экспрессируемого эндогенного гена. В соответствии с вариантом осуществления 12 предложена рекомбинантная последовательность согласно любому из пп. 1-11, где указанная целевая ДНК включает аналитический домен. В соответствии с вариантом осуществления 13 предложена рекомбинантная последовательность согласно любому из пп. 1-12, где указанная целевая ДНК не кодирует пептид. В соответствии с вариантом осуществления 14 предложена рекомбинантная последовательность согласно любому из пп. 1-12, где указанная целевая ДНК кодирует пептид, необязательно кодирующий инсектицидный ген устойчивости, ген невосприимчивости к гербициду, ген эффективности использования азота, ген эффективности использования воды, ген пищевой ценности, ДНК-связывающий ген или селективный маркерный ген. В соответствии с вариантом осуществления 16 предложена рекомбинантная последовательность согласно любому из пп. 1-14, где указанная рекомбинантная последовательность включает следующие характеристики:
a. указанная негенная последовательность содержит меньше 1% метилирования ДНК
b. указанная негенная последовательность демонстрирует частоту рекомбинации от 0,01574 до 83,52 сМ/мпн в геноме сои;
c. указанная негенная последовательность демонстрирует уровень занятости нуклеосомами от 0 до 0,494 генома сои;
d. указанная негенная последовательность обладает менее чем 40% идентичностью последовательности с любой другой последовательностью, содержащейся в геноме сои;
e. указанная негенная последовательность имеет относительное значение местоположения от 0 до 0,99682 отношения геномного расстояния от центромеры хромосомы сои;
f. указанная негенная последовательность имеет процентное содержание гуанина/цитозина в диапазоне от 14,36 до 45,9%;
g. указанная негенная последовательность расположена проксимально к генной последовательности; и,
h. указанная негенная последовательность расположена в 1 мпн области геномной последовательности сои, которая включает одну или более дополнительных негенных последовательностей.
В соответствии с вариантом осуществления 17 предложено растение сои, часть растения сои или клетка растения сои, включающие рекомбинантную последовательность согласно любому из вариантов осуществления 1-14 и 16. В соответствии с вариантом осуществления 18 предложено растение сои, часть растения сои или клетка растения сои согласно варианту осуществления 17, где указанная известная или предсказанная кодирующая последовательность сои экспрессируется на уровне в от 0,000415 до 872,7198. В соответствии с вариантом осуществления 19 предложена рекомбинантная последовательность согласно любому из пп. 1-14, 16 или 17, где указанная целевая ДНК и/или указанная негенная последовательность модифицированы во время вставки указанной целевой ДНК в указанную негенную последовательность.
В соответствии с вариантом осуществления 20 предложен способ создания трансгенной растительной клетки, включающей целевую ДНК, направленную в одну негенную геномную последовательность сои, где способ включает:
a. выбор оптимального негенного геномного локуса сои;
b. введение сайт-специфической нуклеазы в растительную клетку, где сайт-специфическя нуклеаза расщепляет указанную негенную геномную последовательность сои;
c. введение целевой ДНК в растительную клетку;
d. направление целевой ДНК в указанный негенный локус, где расщепление указанной негенной последовательности способствует интеграции полинуклеотидной последовательности в указанный негенный локус; и
e. отбор трансгенных растительных клеток, включающих целевую ДНК, направленную в указанный негенный локус.
ПРИМЕРЫ
Пример 1: Идентификация геномных локусов-мишеней в сое
Геном сои подвергали скринингу с помощью биоинформационного метода, в котором использовали определенные критерии для выбора оптимальных геномных локусов для таргетинга донорного полинуклеотида. Определенные критерии, используемые для выбора геномных локусов, были разработаны с использованием факторов для оптимальной экспрессии трансгена в геноме растения, факторов для оптимального связывания геномной ДНК сайт-специфическим ДНК-связывающим белком и требований к разработке трансгенных растительных продуктов. Для идентификации и выбора геномных локусов наборы геномных и эпигеномных данных генома сои сканировали с использованием биоинформационного метода. Скрининг наборов геномных и эпигеномных данных привел к получению отобранных локусов, которые соответствовали следующим критериям: 1) гипометилированный и с длиной больше 1 тпн; 2) может служить в качестве мишени при опосредованной сайт-специфической нуклеазой интеграции донорного полинуклеотида; 3) агрономически нейтральный или негенный; 4) области, с которых может экспрессироваться интегрированный трансген; и 5) области с рекомбинацией в/около локуса. Соответственно, с использованием указанных определенных критериев идентифицировали в общей сложности 7018 геномных локусов (ID NO:1 SEQ – ID NO:7018). Определенные критерии подробно описаны ниже.
Гипометилирование
Геном сои сканировали для подбора оптимальных геномных локусов длиной больше 1 тпн, которые являлись ДНК гипометилированными. Профили метилирования ДНК в тканях корня и побегов, полученных из Glycine Max сорта Williams82, создавали при использовании метода высокопроизводительного полногеномного секвенирования. Выделенную ДНК подвергали обработке бисульфитом, которая превращает неметилированные цитозины в урацилы, но не воздействует на метилированные цитозины, а затем секвенировали с использованием технологии Illumina HiSeq (Krueger, F. et al. DNA methylome analysis using short bisulfite sequencing data. Nature Methods 9, 145–151 (2012)). Необработанные сиквенсы собирали и картировали в референсном геноме сои сорта Williams82 с использованием программы картирования BismarkTM, как описано в Krueger F, Andrews SR (2011) Bismark: a flexible aligner and methylation caller for Bisulfite-Seq applications. Bioinformatics 27: 1571–1572.
Поскольку в ходе процесса бисульфитной конверсии цитозины в последовательности ДНК, которые метилированы, не превращаются в урацилы, присутствие оснований цитозина в сиквенсах указывают на наличие метилирования ДНК. Считываемые сиквенсы, которые картировали на референсной последовательности, анализировали с целью идентификации геномных положений остатков цитозина с подтверждением метилирования ДНК. Уровень метилирования каждого основания цитозина в геноме вычисляли в процентах от количества метилированных прочтений, картированных в положении определенного основания цитозина, к общему количеству прочтений, картированных в этом положении. Следующее далее предположение объясняет, как уровни метилирования вычисляли для каждого основания в геноме сои. Например, допустим, что основание цитозина находится в положении 100 в хромосоме 1 референсной последовательности сои сорта Williams82. Если в положении 100 есть в общей сложности 20 прочтений, картированных в основание цитозина, и при этом 10 из этих прочтений метилированы, то уровень метилирования для основания цитозина в положении 100 в хромосоме 1 оценивается как 50%. Таким образом, вычисляли профиль уровня метилирования для всех пар оснований геномной ДНК, полученных из ткани корня и побегов сои. Прочтения, которые было нельзя правильно картировать в уникальных положениях в геноме сои, соответствовали повторяющимся последовательностям, которые широко распространены в геноме сои, и, как известно из уровня техники, преимущественно метилированы.
Используя вышеописанную методику, измеряли уровни метилирования генома сои сорта Williams82. Фактически, области генома сои, содержащие метилированные прочтения, указывали, что эти области генома сои были метилированы. Наоборот, области генома сои, в которых отсутствовали метилированные прочтения, указывали, что такие области генома сои не были метилированы. Области генома сои из тканей побегов и корней, которые не были метилированы и не содержали никаких метилированных прочтений, считали "гипометилированными" областями. Чтобы сделать профили метилирования корней и побегов доступными для визуализации, для каждой хромосомы сои сорта Williams82 получали колебательные графики (http://useast.ensembl.org/info/website/upload/wig.html).
После получения уровня метилирования ДНК с разрешением в одну пару оснований в тканях корней и побегов, как описано выше, геном сои подвергали скринингу с использованием окна 100 пн для идентификации геномных областей, которые являются метилированными. Для каждого окна, подвергнутого скринингу в геноме, уровень метилирования ДНК получали при вычислении среднего уровня метилирования каждого основания цитозина в данном окне. Геномные окна с уровнем метилирования ДНК больше 1% называли геномными областями, которые являются метилированными. Метилированные окна, идентифицированные в профилях корней и побегов, комбинировали с получением консенсусного профиля метилирования. Напротив, области в геноме, которые не соответствовали этим критериям и не были идентифицированы как метилированные области в консенсусном профиле, назвали гипометилированными областями. В Таблице 1 представлены идентифицированные гипометилированные области.
Профиль гипометилирования генома сои сорта Williams82.
Эти гипометилированные области генома сои сорта WILLIAMS82 исследовали дополнительно, чтобы идентифицировать и отобрать определенные геномные локусы, поскольку отсутствие метилирования в этих областях указывало на присутствие открытого хроматина. По существу, все последующие исследования проводили на идентифицированных гипометилированных областях.
Пригодность в качестве мишени
Гипометилированные участки, идентифицированные в сое сорта WILLIAMS82, анализировали дополнительно с целью определения, какие участки могут использоваться в качестве мишени при опосредованной сайт-специфической нуклеазой интеграции донорного полинуклеотида. Glycine max, как известно, является палеополиплоидной культурой, которая подвергалась дупликациям генома в своей геномной истории (Jackson et al Genome sequence of the palaeopolyploid soybean, Nature 463, 178-183 (2010)). Геном сои, как известно из уровня техники, содержит длинные отрезки содержащей множество повторов ДНК, которые метилированы и имеют высокие уровни дупликации последовательности. Информация по аннотироанию известных областей с повторами в геноме сои была получена из базы данных генома сои (www.soybase.org, Shoemaker, R.C. et al. SoyBase, the USDA-ARS soybean genetics and genomics database. Nucleic Acids Res. 2010 Jan;38(Database issue):D843-6).
Таким образом, гипометилированные участки, идентифицированные выше, подвергали скринингу с целью удаления любых участков, которые выравниваются с известными областями, содержащими повторы, аннотированными в геноме сои. Оставшиеся гипометилированные участки, которые прошли этот первый скрининг, сканировали далее с использованием поиска гомологии на основе BLASTTM в геномной базе данных сои с помощью программы NCBI BLASTTM+ (версия 2.2.25), которую запускали при использовании параметров по умолчанию (Stephen F. Altschul et al (1997) Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res. 25:3389-3402). В результате скрининга BLASTTM любые гипометилированные участки, которые имели значительные совпадения в других областях в геноме, с охватом выравнивания последовательности более 40%, исключали из дальнейших исследований.
Агрономически нейтральный или негенный
Гипометилированные участки, идентифицированные в сое сорта William82, анализированы далее с целью определения, какие участки являлись агрономически нейтральными или негенными. По существу, гипометилированные участки, описанные выше, подвергали скринингу для удаления любых участков, которые перекрывались или содержали любые известные или предсказанные эндогенные кодирующие последовательности сои сорта William82. С этой целью данные по аннотированию известных генов и информацию по картированию маркерных экспрессируемых последовательностей (EST) получали из геномной базы данных сои (www.soybase.org - использовали генные модели версии 1.1, Jackson et al Genome sequence of the palaeopolyploid soybean Nature 463, 178-183 (2010)). Также рассматривали любую геномную область непосредственно 2 тпн до и 1 тпн после открытой рамки считывания. Такие 5' и 3' области могут содержать известные или неизвестные консервативные регуляторные элементы, которые являются существенными для функции гена. Гипометилированные участки, описанные выше, анализировали на предмет присутствия известных генов (включая 2 тпн области 5' и 1 тпн области 3') и EST. Любые гипометилированные участки, которые выравнивались или перекрывались с известными генами (включая 2 тпн области 5' и 1 тпн области 3') или EST, исключали из последующего анализа.
Экспрессия
Гипометилированные участки, идентифицированные в сое сорта Williams82, анализированы далее с целью определения, какие участки находились вблизи от экспрессируемого гена сои. Уровень экспрессии транскриптов генов сои измеряли, анализируя данные профилирования транскриптома, полученные из тканей корней и побегов сои сорта Williams82 при использовании технологии RNAseqTM, как описано в Mortazavi et al., Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nat Methods. 2008; 5(7):621–628, и Shoemaker RC et al., RNA-Seq Atlas of Glycine max: a guide to the soybean Transcriptome. BMC Plant Biol. 2010 Aug 5; 10:160. Для каждого гипометилированного участка выполняли анализ с целью идентификации любого аннотированного гена, присутствующего в 40 тпн области вблизи от гипометилированного участка, а также среднего уровня экспрессии аннотированного гена(ов), расположенного вблизи от гипометилированного участка. Гипометилированные участки, расположенные дальше 40 тпн от аннотированного гена с ненулевым средним уровнем экспрессии были определены как не являющиеся проксимальными к экспрессируемому гену сои и были исключены от последующих исследований.
Рекомбинация
Гипометилированные участки, идентифицированные в сое сорта Williams82, анализировали далее с целью определения, какие участки имели подтверждение рекомбинации и могли способствовать интрогрессии оптимальных геномных локусов в другие линии сои путем обычного скрещивания. Различные генотипы сои обычно скрещивают в процессе обычной селекции с получением новых и улучшенных линий сои, содержащих признаки, представляющие агрономический интерес. Также, агрономические признаки, которые интрогрессируют в оптимальные геномные локусы в линии сои посредством растение-опосредованной трансформации трансгена, должны быть способными к последующей интрогрессии в другие линии сои, в особенности элитные линии, посредством мейотической рекомбинации в процессе обычного скрещивания растений. Гипометилированные участки, описанные выше, подвергали скринингу с целью идентификации и отбора участков, которые обладали некоторым уровнем мейотической рекомбинации. Любые гипометилированные участки, которые присутствовали в хромосомных областях, характеризуемые как "холодные точки" рекомбинации, идентифицировали и исключали. В сое такие холодные точки определяли при использовании набора маркерных данных, полученного с рекомбинантной инбредной картирующей популяцией (Williams 82 x PI479752). Этот набор данных состоял из ~16600 SNP маркеров, которые могли быть физически картированы на референсной геномной последовательности Glycine max.
Частоты мейотической рекомбинации между любой парой геномных маркеров сои по хромосоме вычисляли на основе отношения генетического расстояния между маркерами (в сантиморганах (сМ)) к физическому расстоянию между маркерами (в мегабазах (мпн)). Например, если генетическое расстояние между парой маркеров составляло 1 сМ, а физическое расстояние между той же парой маркеров составляло 2 мпн, то определяемая расчетная частота рекомбинации составляла 0,5 сМ/мпн. Для каждого гипометилированного участка, идентифицированного выше, выбирали пару маркеров, по меньшей мере с интервалом 1 мпн, и вычисляли частоту рекомбинации. Развитие этого способа использовали для вычисления частоты рекомбинации гипометилированных участков. Любые гипометилированные участки с частотой рекомбинации 0 сМ/мпн идентифицировали и исключали из дальнейшего анализа. Оставшиеся гипометилированные области, включающие частоту рекомбинации больше 0 сМ/мпн, отбирали для последующего анализа.
Идентификация оптимальных геномных локусов
Применение критериев отбора, описанных выше, привело к идентификации в общей сложности 90325 оптимальных геномных локусов в геноме сои. В Таблице 2 представлены длины идентифицированных оптимальных геномных локусов. Эти оптимальные геномные локусы обладают следующими характеристиками: 1) гипометилированные геномные локусы имеют длину больше 1 тпн; 2) геномные локусы, которые являются мишенями при опосредованной сайт-специфической нуклеазой интеграции донорного полинуклеотида; 3) геномные локусы, которые являются агрономически нейтральными или негенными; 4) геномные локусы, с которых может экспрессироваться трансген; и 5) подтверждение рекомбинации в геномных локусах. Из всех оптимальных геномных локусов, описанных в Таблице 2, далее анализировали и использовали для таргетинга донорной полинуклеотидной последовательности только оптимальные геномные локусы, которые имели длину больше 1 тпн. Последовательности этих оптимальных геномных локусов раскрыты в SEQ ID NO:1–SEQ ID NO:7018. В совокупности эти оптимальные геномные локусы являются положениями в геноме сои, в которые может быть направлена донорная полинуклеотидная последовательность, как продемонстрировано ниже.
Перечислен размер оптимальных геномных локусов, идентифицированных в геноме сои, которые являются гипометилированными, демонстрируют подтверждение рекомбинации, могут служить в качестве мишеней, являются агрономически нейтральными или негенными и находятся вблизи от экспрессируемого эндогенного гена.
Пример 2: F-распределение и анализ главных компонент для кластеризации оптимальных геномных локусов из сои
7018 идентифицированных оптимальных геномных локусов (SEQ ID NO: 1-SEQ ID NO: 7018) анализировали далее при использовании статистических методов F-распределения и анализа главных компонент для определения репрезентативной совокупности и кластеров для группировки оптимальных геномных локусов.
Анализ F-распределения
7018 идентифицированных оптимальных геномных локусов анализировали статистически при использовании статистического анализа непрерывного распределения вероятности. В качестве варианта статистического анализа непрерывного распределения вероятности выполняли тест F-распределения для определения репрезентативного количества оптимальных геномных локусов. Анализ с использованием теста F-распределения выполняли, используя уравнения и методы, известные специалистам. Для получения дополнительных сведений, анализ с использованием теста F-распределения, как описано в статье K.M Remund, D. Dixon, DL. Wright and LR. Holden. Statistical considerations in seed purity testing for transgenic traits. Seed Science Research (2001) 11, 101–119, включенной в настоящую заявку посредством отсылки, является неограничивающим примером теста F-распределения. Тест F-распределения предполагает случайную выборку оптимальных геномных локусов, при этом любые недействительные локусы были равномерно распределены среди 7018 оптимальных геномных локусов, и количество оптимальных геномных выбранных локусов составляет 10% или меньше от общей совокупности 7018 оптимальных геномных локусов.
Анализ F-распределения показал, что 32 из 7018 оптимальных геномных локусов обеспечивали репрезентативное количество 7018 оптимальных геномных локусов, при 95% доверительном уровне. Таким образом, анализ F-распределения показал, что если бы были протестированы 32 оптимальных геномных локуса, и во все могла быть направлена донорная полинуклеотидная последовательность, то эти результаты показали бы, что 91 или больше из 7018 оптимальных геномных локусов являются положительными при 95% доверительном уровне. Наилучшая оценка подтверждения общего процента от 7018 оптимальных геномных локусов была бы в случае, если бы 100% из 32 протестированных оптимальных геномных локусов могли быть мишенями. Соответственно, 91% фактически является нижней границей истинного процента, подтвержденного при 95% доверительном уровне. Эта нижняя граница основана на 0,95 квантиле F-распределения при 95% доверительном уровне (Remund K, Dixon D, Wright D, and Holden L. Statistical considerations in seed purity testing for transgenic traits. Seed Science Research (2001) 11, 101–119).
Анализ главных компонент
Следующим этапом применяли статистический метод анализа главных компонент (PCA) для дополнительной оценки и визуализации сходств и различий набора данных, включающего 7018 идентифицированных оптимальных геномных локусов, чтобы обеспечить выборку различных локусов для проверки подтверждения таргетинга. PCA включает математический алгоритм, который преобразует большее количество коррелированных переменных в меньшее количество некоррелированных переменных, названных главными компонентами.
PCA выполнили на 7018 идентифицированных оптимальных геномных локусах, получив набор вычисляемых параметров или признаков, которые могли использоваться для описания 7018 идентифицированных оптимальных геномных локусов. Каждый параметр может быть вычислен численно и специфически определен с получением геномного и эпигеномного контекста 7018 идентифицированных оптимальных геномных локусов. Набор из 10 параметров для каждого из оптимальных геномных локусов сои был идентифицирован и описан более подробно ниже.
1. Длина оптимальных геномных локусов
a. Длина оптимальных геномных локусов в этом наборе данных варьировала от минимум 1000 пн до максимум 5713 пн.
2. Частота рекомбинации в 1 мпн области вокруг оптимальных геномных локусов
a. В сое частоту рекомбинации для хромосомного положения определяли при использовании внутреннего набора маркерных данных высокого разрешения, полученного из множеств картирующих популяций.
b. Частоты рекомбинации между любыми парами маркеров на хромосоме вычисляли на основе отношения генетического расстояния между маркерами (в сантиморганах (сМ)) к физическому расстоянию между маркерами (в мпн). Например, если генетическое расстояние между парой маркеров составляет 1 сМ, а физическое расстояние между теми же парами маркеров составляет 2 мпн, расчетная частота рекомбинации составляет 0,5 сМ/мпн. Для каждого оптимального геномного локуса была выбрана пара маркеров с интервалом по меньшей мере 1 МБ, и таким образом была вычислена частота рекомбинации. Эти значения рекомбинации варьировали от минимум 0,01574 сМ/мпн до максимум 83,52 сМ/мпн.
3. Уровень уникальности последовательности оптимальных геномных локусов
a. Для каждого из оптимальных геномных локусов нуклеотидную последовательность оптимальных геномных локусов сканировали в геноме сои сорта Williams82, используя поиск гомологии на основе BLASTTM, при использовании программы NCBI BLASTTM+ (версия 2.2.25), которую запускали с параметрами по умолчанию (Stephen F. Altschul et al (1997), "Gapped BLAST and PSI-BLAST: a new generation of protein database search programs", Nucleic Acids Res. 25:3389-3402). При идентификации последовательности этих оптимальных геномных локусов в геноме сои сорта Williams82, первое совпадение BLASTTM, идентифицированное с помощью данного поиска, представляет последовательность сои сорта Williams82 непосредственно. Идентифицировали второе совпадение BLASTTM для каждой последовательности оптимальных геномных локусов, и охват выравнивания (представленный как процент оптимальных геномных локусов, покрытых совпадение BLASTTM) совпадения использовали в качестве показателя уникальности последовательности оптимальных геномных локусов в геноме сои. Эти значения охвата выравнивания для второго совпадения BLASTTM варьировали от минимум 0% до максимум 39,97% идентичности последовательности. Любые последовательности, которые выравнивались с более высокими уровнями идентичности последовательности, не рассматривали.
4. Расстояние от оптимальных геномных локусов до ближайшего соседнего гена
a. Информацию по аннотированию и местоположению известных генов в геноме сои получали из Базы данных генома сои (доступной на сайте www.soybase.org - использовались генные модели версии 1.1, Jackson et al Genome sequence of the palaeopolyploid soybean, Nature 463, 178-183 (2010)). Для каждого из оптимальных геномных локусов идентифицировали ближайший аннотированный ген, рассматривая положения до и после них, и измеряли расстояние между последовательностью оптимальных геномных локусов и геном (в пн). Например, если оптимальный геномный локус расположен на хромосоме Gm01 с положения 2500 до положения 3500, а ближайший ген к этому оптимальному геномному локусу расположен на хромосоме Gm01 с положения 5000 до положения 6000, вычисленное расстояние от оптимальных геномных локусов до этого ближайшего гена составлят 1500 пн. Эти значения для набора данных всех 7018 оптимальных геномных локусов варьировали от минимум 1001 пн до максимум 39482 пн.
5. % GC в последовательности оптимальных геномных локусов
a. Для каждого оптимального геномного локуса анализировали нуклеотидную последовательность для оценки количества присутствующих оснований гуанина и цитозина. Это значение было представлено в виде процента от длины последовательности каждого оптимального геномного локуса и дает показатель % GC. Эти значения % GC для набора данных оптимальных геномных локусов сои варьировали от 14,4% до 45,9%.
6. Количество генов в 40 тпн области, окружающей последовательность оптимальных геномных локусов
a. Информацию по аннотированию и местоположению известных генов в геноме сои сорта Williams82 получали из Базы данных генома сои. Для каждой из 7018 последовательностей оптимальных геномных локусов определяли окно протяженностью 40 тпн вокруг последовательности оптимальных геномных локусов и подсчитывали количество аннотированных генов, положения которых накладывались на это окно. Эти значения варьировали от минимум 1 гена до максимум 18 генов в окружающей 40 тпн области.
7. Средняя экспрессия гена в 40 тпн области вокруг оптимальных геномных локусов
a. Уровень экспрессии транскриптов генов сои измеряли с помощью анализа доступных данных профилирования транскриптома, полученных из тканей корней и побегов сои сорта Williams82 при использовании технологии RNAseqTM. Информацию по аннотированию и местоположению известных генов в геноме сои сорта Williams82 получали из Базы данных генома сои. Для каждого оптимального геномного локуса идентифицировали аннотированные гены в геноме сои сорта Williams82, которые присутствовали в 40 тпн области вокруг оптимальных геномных локусов. Уровни экспрессии для каждого из генов получали из профилей транскриптома, описанных в указанных выше ссылках, и вычисляли средний уровень экспрессии генов. Значения экспрессии всех генов в геноме сои существенно различаются. Значения средней экспрессии для набора данных всех 7018 оптимальных геномных локусов варьировали от минимум 0,000415 до максимум 872,7198.
8. Уровень занятости нуклеосомами вокруг оптимальных геномных локусов
a. Понимание уровня занятости нуклеосомами для конкретной нуклеотидной последовательности дает информацию о хромосомных функциях и геномном контексте последовательности. Статистический пакет NuPoPTM использовали для предсказания занятости нуклеосомами и создания карты наиболее вероятного расположения нуклеосом для геномных последовательностей любого размера (Xi, L., Fondufe-Mittendor, Y., Xia, L., Flatow, J., Widom, J. and Wang, J.-P., Predicting nucleosome positioning using a duration Hidden Markov Model, BMC Bioinformatics, 2010, doi:10.1186/1471-2105-11-346). Для каждого из 7018 оптимальных геномных локусов нуклеотидная последовательность была представлена для анализа с использованием программы NuPoPTM, при этом вычисляли показатель занятости нуклеосомами. Эти показатели занятости нуклеосомами для набора данных оптимальных геномных локусов сои варьировали от минимум 0 до максимум 0,494.
9. Относительное положение на хромосоме (близость к центромере)
a. Центромера представляет собой область на хромосоме, которая соединяет две сестринских хроматиды. Части хромосомы с обеих сторон центромеры известны как плечи хромосомы. Геномные положения центромер на всех 20 хромосомах сои были идентифицированы в опубликованной референсной последовательности сои сорта Williams82 (Jackson et al Genome sequence of the palaeopolyploid soybean Nature 463, 178-183 (2010)). Информация о положении центромеры в каждой из хромосом сои и длинах плечей хромосомы была получена из базы данных генома сои. Для каждого оптимального геномного локуса измеряли (в пн) геномное расстояние от последовательности оптимального геномного локуса до центромеры хромосомы, на которой он расположен. Относительное положение оптимальных геномных локусов на хромосоме представлено как отношение их геномного расстояния до центромеры к длине определенного хромосомного плеча, на котором он находится. Эти значения относительного местоположения для набора данных оптимальных геномных локусов сои варьировали от минимум 0 до максимум 0,99682 отношения геномного расстояния.
10. Количество оптимальных геномных локусов в 1 мпн области
a. Для каждого из оптимальных геномных локусов определяли геномное окно протяженностью 1 мпн вокруг местоположения оптимальных геномных локусов и вычисляли количество других, дополнительных оптимальных геномных локусов, присутствующих в этой области или накладывающихся на нее, включая рассматриваемые оптимальные геномные локусы. Количество оптимальных геномных локусов в 1 мпн варьировало от минимум 1 до максимум 49.
Все 7018 оптимальных геномных локусов были проанализированы с использованием параметров и признаков, описанных выше. Результаты или значения для оценки параметров и признаков каждого оптимального геномного локуса описаны далее в Таблице 3 (в настоящей заявке включена посредством отсылки как отдельный электронный файл). Полученный в результате набор данных использовали в статистическом методе PCA для разделения 7018 идентифицированных оптимальных геномных локусов на кластеры. Во время процесса кластеризации, после оценки "p" главных компонент оптимальных геномных локусов, распределение оптимальных геномных локусов в один из 32 кластеров продолжалось в "p"-мерном евклидовом пространстве. Каждую из "p" осей разбивали на "k" интервалов. Оптимальные геномные локусы, распределенные в один интервал, группировали вместе с образованием кластеров. Используя этот анализ, каждую ось PCA разбивали на два интервала, который выбирали на основе априорной информации относительно количества кластеров, требуемых для экспериментального подтверждения. Весь анализ и визуализацию полученных кластеров выполняли с помощью программы Molecular Operating EnvironmentTM (MOE), выпущенной Chemical Computing Group Inc. (Montreal, Quebec, Canada).
Метод PCA использовали для разделения набора из 7018 идентифицированных оптимальных геномных локусов на 32 различных кластера на основе значений их параметров, описанных выше. Во время процесса PCA получили пять главных компонент (ПК), при этом три лучших PC содержали приблизительно 90% общей вариации в наборе данных (Таблица 4). Эти три PCA использовали для графического представления 32 кластеров на трехмерном графике (Фиг. 1). После завершения процесса кластеризации из каждого кластера выбрали один репрезентативный оптимальный геномный локус. Это было выполнено посредством выбора из каждого кластера отобранного оптимального геномного локуса, который был наиболее близок к центроиду данного кластера (Таблица 4). Хромосомные положения 32 репрезентативных оптимальных геномных локусов однородно распределены по 20 хромосомам сои и не смещены ни к одному специфическому геномному положению, как показано на Фиг. 2.
Описание 32 репрезентативных оптимальных геномных локусов сои, идентифицированных в результате PCA
Конечный выбор геномных локусов для таргетинга донорной полинуклеотидной последовательности
В общей сложности 32 геномных локуса были идентифицированы и отобраны для таргетинга донорной полинуклеотидной последовательности из 7018 геномных локусов, которые были разделены на 32 различных кластера. Для каждой из 32 кластеров был выбран репрезентативный геномный локус (ближайший к центроиду кластера, как описано выше в Таблице 4) или дополнительный локус с гомологией к линию таргетинга. Дополнительные оптимальные геномные локусы были выбраны при первом скрининге всех 7018 выбранных оптимальных геномных последовательностей в базы данных полных геномов, состоящей из данных геномных последовательностей ДНК для Glycine max сорта Maverick (линии трансформации и скрининга таргетинга) и Glycine max сорта Williams82 (референсная линия) с целью определения охвата (сколько оптимальных геномных локусов присутствовало в обоих геномах) и процента идентичности последовательностей в геноме обеих линий. Оптимальные геномные локусы со 100% охватом (выравниванием всей длины последовательности оптимальных локусов между обоими геномами) и 100% идентичностью в базах геномных данных Williams82 были выбраны для подтверждения таргетинга. Другие критерии, такие как размер геномных локусов, степень уникальности, % содержание GC и хромосомное распределение оптимальных геномных локусов также учитывали при выборе дополнительных оптимальных геномных локусов. Хромосомное положение 32 выбранных оптимальных геномных локусов и определенная геномная конфигурации каждого из оптимальных геномных локусов сои показаны на Фиг. 3 и в Таблице 5, соответственно.
Описание 32 выбранных оптимальных геномных локусов сои, отобранных для проверки подтверждения таргетинга. Из этих оптимальных геномных локусов, представленных в этой таблице, пример расщепления и таргетинга 32 оптимальных геномных локусов сои является репрезентативным для всех идентифицированных 7018 выбранных оптимальных геномных локусов сои.
Большой набор из 7018 геномных положений идентифицировали в геноме сои в качестве оптимальных геномных локусов для таргетинга донорной полинуклеотидной последовательности с применением технологии точной геномной инженерии. Метод статистического анализа применили для разделения 7018 выбранных геномных локусов на 32 кластера со сходным геномным контекстом и идентификации подгруппы из 32 выбранных геномных локусов, репрезентативных для набора из 7018 выбранных геномных локусов. 32 репрезентативных локуса подтверждали в качестве оптимальных геномных локусов посредством таргетинга донорной полинуклеотидной последовательности. При выполнении статистического анализа PCA для числовых значений, полученных для десяти наборов параметров или признаков, которые описаны выше, десять параметров или признаки преобразовали в компоненты PCA меньшей размерности. Фактически, компоненты PCA разделили на пять показателей, которые являются репрезентативными для десяти параметров или признаков, описанных выше (Таблица 6). Каждый компонент PCA эквивалентен комбинации из десяти параметров или признаков, описанных выше. Из этих компонентов PCA, составляющих пять показателей, вычисленных с использованием статистического анализа PCA, были определены 32 кластера.
Пять компонент РСА (PCA1, PCA2, PCA3, PCA3,PCA4 и PCA5), которые определяют каждый из 32 кластеров, и последовательности (ID NO:1 SEQ-SEQ ID NO: 7018), которые составляют каждый кластер. Эти пять показателей являются репрезентативными для десяти параметров или признаков, описанных выше, которые использовали для идентификации оптимальных геномных локусов. Приведены минимальные (Мин), средние, срединные и максимальные (Макс) значения для каждой компоненты РСА.
(SEQ ID NO:3676 -- SEQ ID NO:3992)
(SEQ ID NO: 4888-- SEQ ID NO:5062)
Пример 3: Конструкция цинковых пальцев для связывания геномных локусов в сое
Цинк-пальцевые белки, направленные против идентифицированных последовательностей ДНК репрезентативных геномных локусов, были сконструированы, как описано ранее. См., например, Urnov et al., (2005) Nature 435:646-551. Примерные последовательности-мишени и спирали распознавания показаны в Таблице 7 (конструкции областей спиралей распознавания) и Таблице 8 (сайты-мишени). В Таблице 8 нуклеотиды в сайте-мишени, с которыми контактируют спирали распознавания ZFP, показаны заглавными буквами, а нуклеотиды, с которыми они не контактируют, показаны строчными буквами. Сайты-мишени цинк-пальцевой нуклеазы (ZFN) были сконструированы для всех ранее описанных 32 выбранных оптимальных геномных локусов. Множество конструкций ZFP разработали и протестировали с целью идентификации пальцев, которые связываются с наибольшей эффективностью с различными сайтами-мишенями в 32 репрезентативных геномных локусах, которые были идентифицированы и отбраны в сое, как описано выше. Определенные спирали распознавания ZFP (Таблица 7), которые связываются с наибольшей эффективностью с последовательностями распознавания цинковых пальцев, использовали для таргетинга и интеграции донорной последовательности в геном сои.
Конструкции цинковых пальцев для выбранных геномных локусов сои (N/A обозначает "не применим").
Сайт-мишень цинкового пальца в выбранных геномных локусах сои
TACTTGCTGGTA
ATCATCTGCAAA
Конструкции цинковых пальцев репрезентативных геномных локусов сои включали в векторы экспрессии цинковых пальцев, кодирующие белок, имеющий по меньшей мере один палец со структурой CCHC. См., патентную публикацию США 2008/0182332. В частности, последний палец в каждом белке имеет каркас CCHC для спирали распознавания. Неканонические кодирующие последовательности цинковых пальцев были слиты с нуклеазным доменом рестриктазы типа IIS FokI (аминокислоты 384-579 в последовательности Wah et al., (1998) Proc. Natl. Acad. Sci. USA 95:10564-10569) через ZC линкер из четырех аминокислот и сигналом ядерной локализации opaque-2, оптимизированным для сои, с получением цинк-пальцевых нуклеаз (ZFN). См. патент США 7,888,121. Цинковые пальцы для различных функциональных доменов были отобраны для применения in vivo. Из множества ZFN, которые были сконструированы, получены и протестированы на связывание с предполагаемым геномным сайтом-мишенью, ZFN, описанные в Таблице 8 выше, были идентифицированы как обладающие активностью in vivo и были охарактеризованы как способные к эффективному связыванию и расщеплению уникальных полинуклеотидных геномных сайтов-мишеней сои in planta.
Сборка конструкций ZFN
Плазмидные векторы, содержащие экспрессионные конструкции гена ZFN, были сконструированы и получены при использовании навыков и методик, общеизвестных в уровне техники (см., например, Ausubel или Maniatis). Каждая ZFN-кодирующая последовательность была слита с последовательностью, кодирующей сигнал ядерной локализации opaque-2 (Maddaloni et al., (1989) Nuc. Acids Res. 17:7532), который был расположен перед цинк-пальцевой нуклеазой. Неканонические последовательности, кодирующие цинк-пальцевые нуклеазы, были слиты с нуклеазным доменом рестриктазы типа IIS FokI (аминокислоты 384-579 в последовательности из Wah et al. (1998) Proc. Natl. Acad. Sci. USA 95:10564-10569). Экспрессию слитых белков направлял сильный конститутивный промотор из вируса мозаики жилок маниока. Кассета экспрессии также включала 3'-UTR из ORF23 Agrobacterium tumefaciens. Автогидролизующийся 2A, кодирующий нуклеотидную последовательность из вируса Thosea asigna (Szymczak et al., (2004) Nat Biotechnol. 22:760-760), добавляли между двумя слитыми белками цинк-пальцевых нуклеаз, которые клонировали в конструкцию.
Плазмидные векторы собирали при использовании технологии IN-FUSIONTM Advantage (Clontech, Mountain View, CA). Эндонуклеазы рестрикции были получены от New England BioLabs (Ipswich, MA), и ДНК лигазу T4 (Invitrogen, Carlsbad, CA) использовали для лигирования ДНК. Получение плазмид выполняли при использовании набора NUCLEOSPIN® Plasmid Kit (Macherey-Nagel Inc., Bethlehem, PA) или Plasmid Midi Kit (Qiagen) в соответствии с инструкциями поставщиков. Фрагменты ДНК выделяли при использовании набора для выделения из геля QIAquick Gel Extraction KitTM (Qiagen) после электрофореза в агарозном геле с трис-ацетатным буфером. Колонии всех реакций лигирования подвергали первичному скринингу путем рестрикции минипрепарата ДНК. Плазмидную ДНК отобранных клонов секвенировали с помощью поставщика коммерческих услуг секвенирования (Eurofins MWG Operon, Huntsville, AL). Данные последовательностей были собраны и проанализированы с использованием программы SEQUENCHERTM (Gene Codes Corp., Ann Arbor, MI). Плазмиды были сконструированы и проверены с помощью рестрикионного анализа и секвенирования ДНК.
Клонирование цинковых пальцев в автоматизированном процессе
Подгруппу векторов цинк-пальцевых нуклеаз клонировали с помощью автоматизированной линии для конструирования ДНК. В общем, автоматизированная линия позволила получить векторные конструкции с идентичной архитектурой ZFN, как описано ранее. Каждый мономер цинкового пальца, который придает ДНК-связывающую специфичность ZFN, был разделен на 2-3 уникальных последовательности в аминокислотном мотиве KPF. На 5’ и 3’ концах фрагменты ZFN модифицировали включением сайта распознавания BsaI (GGTCTCN) и соответствующими липкими концами. Липкие концы были распределены таким образом, чтобы сборка 6-8 частей давала только требуемый полноразмерный экспрессионный клон. Модифицированные ДНК фрагменты синтезировали de novo (Synthetic Genomics Incorporated, La Jolla, CA). Одну основу для двудольных растений, pDAB118796, использовали во всех конструкциях ZFN сои. Он содержал промотор из вируса мозаики маниока и NLS Opaque2, а также домен FokI и 3’UTR Orf23 из Agrobacterium tumefaciens. Между NLS Opaque 2 и доменом FokI клонировали фланкированный сайтами BsaI ген SacB из Bacillus subtilis. При посеве предполагаемых продуктов лигирования на чашки со средой, содержащей сахарозу, кассета SacB действовала в качестве агента негативной селекции, снижающего или исключающего контаминацию векторной основы. Другой частью, неоднократно используемой во всех конструкциях, являлся pDAB117443. Этот вектор содержит первый мономер домен Fok1, прерывающую последовательность T2A и 2-ой мономер NLS Opaque2, все фланкированы сайтами BsaI.
Используя эти материалы в качестве библиотеки ДНК-фрагментов ZFN, станция Freedom Evo 150® (TECAN, Mannedorf, Switzerland) производила добавление по 75-100 нг каждой ДНК плазмиды или синтезированного фрагмента из пробирок с двумерным штриховым кодом в ПЦР-планшет (ThermoFisher, Waltham, MA). К реакции добавляли BsaI (NEB, Ipswich, MA) и ДНК-лигазу T4 (NEB, Ipswich, MA) с добавкой белка бычьего сывороточного альбумина (NEB, Ipswich, MA) и буфера для ДНК-лигазы T4 (NEB, Ipswich, MA). Реакции проводили циклами (25×) с инкубированием в течение 3 минут при 37°C и 4 минут при 16°C в амплификаторе C1000 Touch Thermo Cycler® (BioRad, Hercules, CA). Лигированный материал трансформировали и подвергали скринингу в Top10 cells® (Life Technologies Carlsbad, CA) вручную или при использовании системы отбора колоний Qpix460 и LabChip GX® (Elmer, Waltham, MA). Правильно расщепляемые колонии подверждали по последовательности и использовали для трансформации растений.
Сборка универсальной донорной конструкции
Для обеспечения быстрого тестирования большого количества локусов-мишеней была разработана и создана новая, гибкая система универсальной донорной последовательности. Универсальная донорная полинуклеотидная последовательность была совместима с высокопроизводительными методами конструирования и анализа векторов. Универсальная донорная система состояла по меньшей мере из трех модульных доменов: вариабельного ZFN-связывающего домена, аналитического невариабельного домена и домена с заданными пользователем параметрами, а также простой плазмидной основы для масштабирования вектора. Невариабельная универсальная донорная полинуклеотидная последовательность была общей для всех доноров и позволяла применять схему с конечным набором анализов, которые могут использоваться для всех сайтов-мишеней в сое, с обеспечением, таким образом, единообразия при оценке таргетинга и уменьшения продолжительности аналитического цикла. Модульная природа этих доменов позволяет проводить высокопроизводительную сборку донора. Кроме того, универсальная донорная полинуклеотидная последовательность обладает другими уникальными особенностями, которые направлены на упрощение последующего анализа и совершенствование интерпретации результатов. Она содержит асимметричную последовательность сайта рестрикции, которая позволяет выполнять расщепление ПЦР-продуктов с получением фрагментов требуемого размера. Последовательности, содержащие вторичные структуры, которые могли мешать проведению ПЦР-амплификации, удаляли. Универсальная донорная полинуклеотидная последовательность имела малый размер (меньше 3,0 тпн). Наконец, универсальная донорная полинуклеотидная последовательность была сконструирована с применением высококопийной основы pUC19, которая обеспечивает быстрое накопление большого количества тестируемой ДНК.
В качестве варианта осуществления пример плазмиды, включающей универсальную донорную полинуклеотидную последовательность, представлен как pDAB124280 (SEQ ID NO:7561 и Фигура 7). В дополнительном варианте осуществления универсальная донорная полинуклеотидная последовательность представлена как: pDAB124281, SEQ ID NO:7562, Фигура 8; pDAB121278, SEQ ID NO:7563, Фигура 9; pDAB123812, SEQ ID NO:7564, Фигура 10; pDAB121937, SEQ ID NO:7565, Фигура 11; pDAB123811, SEQ ID NO:7566, Фигура 12; и pDAB124864, SEQ ID NO:7567, Фигура 13. В другом варианте осуществления могут быть сконструированы дополнительные последовательности, включающие универсальную донорную полинуклеотидную последовательность с функционально экспрессируемой кодирующей последовательностью или нефункциональными (беспромоторными) экспрессируемыми кодирующими последовательностями (Таблица 11).
Представлены различные последовательности универсальных доменов, которыми трансформировали протопласты растительных клеток для донор-опосредованной интеграции в геном сои. Различные элементы системы плазмид с универсальными доменами описаны и идентифицированы положением пар оснований в прилагаемом SEQ ID NO:. "N/A" означает не применим.
Универсальная донорная полинуклеотидная последовательность представляла собой малую модульную донорную систему размером 2-3 тпн, доставляемую в виде плазмиды. Она являлась минимальным донором, включающим 1, 2, 3, 4, 5, 6, 7, 8, 9 или больше сайтов связывания ZFN, короткую 100-150 пн матричную область, называемую "ДНК X" или "последовательность UZI" (SEQ ID NO:7568), которая несет сайты рестрикции и последовательности ДНК для подбора праймеров или кодирующие последовательности, и простую основу плазмиды (Фиг. 4). Вся плазмида была вставлена посредством NHEJ после введения двухцепочечных разрывов ДНК по подходящему сайту связывания ZFN; сайты связывания ZFN могут быть включены тандемно. Такой вариант осуществления универсальной донорной полинуклеотидной последовательности являлся наиболее подходящим для быстрого скрининга сайтов-мишеней и ZFN, при этом последовательности, которые было сложно амплифицировать, были минимизированы в доноре. Также были созданы универсальные доноры без последовательности "UZI", но несущие один или более сайтов ZFN.
В другом варианте осуществления универсальная донорная полинуклеотидная последовательность состояла по меньшей мере из 4 модулей и несла связывающие сайты ZFN, плечи гомологии, ДНК X либо только с приблизительно 100 пн аналитической частью, либо с кодирующими последовательностями. Этот вариант универсальной донорной полинуклеотидной последовательности подходил для исследования HDR-опосредованной вставки гена во множестве сайтов-мишеней, с использованием нескольких ZFN (Фиг. 5).
Универсальная донорная полинуклеотидная последовательность может применяться со всеми направляющими молекулами с определенными ДНК-связывающими доменами, с двумя механизмами направленной донорной вставки (NHEJ/HDR). По существу, в случае, когда универсальная донорная полинуклеотидная последовательность была совместно введена с подходящей ZFN экспрессионной конструкцией, донорный вектор и геном сои были разрезаны в одном определенном положении, которое определяется связыванием специфической ZFN. После линеаризации донор может быть включен в геном посредством NHEJ или HDR. Различные аналитические особенности при создании вектора могут быть также применены для определения цинкового пальца, который максимально повышает эффективность доставки при направленной интеграции.
Пример 4: Методики трансформации сои
Перед доставкой в протопласты Glycine max сорта Maverick, плазмидную ДНК для каждой ZFN конструкции получали из культур E. coli при использовании Pure Yield Plasmid Maxiprep System® (Promega Corporation, Madison, WI) или Plasmid Maxi Kit® (Qiagen, Valencia, CA) согласно инструкциям поставщиков.
Выделение протопластов
Протопласты выделяли из суспензионной культуры Maverick, полученной из каллуса, полученного из эксплантов листьев. Суспензии субкультивировали каждые 7 дней в свежей среде LS (Linsmaier and Skoog 1965), содержащей 3% (в/об) сахарозы, 0,5 мг/л 2,4-D и 7 г бактоагара, pH 5,7. Для выделения тридцать миллилитров суспензионной культуры Maverick через 7 дней субкультивирования переносили в коническую пробирку объемом 50 мл и центрифугировали при 200 g в течение 3 минут с получением приблизительно 10 мл объема осажденных клеток (SCV) в пробирке. Супернатант удаляли и добавляли двадцать миллилитров раствора ферментов (0,3% пектолиазы (320952; MP Biomedicals), 3% целлюлазы ("Onozuka" R10TM; Yakult Pharmaceuticals, Japan) в растворе MMG (4 мМ MES, 0,6 М маннита, 15 мМ MgCl2, pH 6,0) на каждые 4 SCV суспензионных клеток и оборачивали пробирки пленкой ParafilmTM. Пробирки устанавливали в шейкер на ночь (приблизительно на 16-18 ч) и аликвоту обработанных ферментами клеток тщательно исследовали под микроскопом, чтобы гарантировать, что расщепление клеточной стенки являлось достаточным.
Очистка протопластов
Метод трансформации на основе протопластов сои (Glycine max сорта Maverick) разработали и использовали для трансформации протопластов сои. Протопласты выделяли из суспензионной культуры Maverick, полученной из каллуса, полученного из эксплантов листьев. Методики, представленные ниже, описывают данный метод. Суспензии клеток сои субкультивировали каждые 7 дней при разведении 1:5 в свежей среде LS (Linsmaier and Skoog 1965), содержащей 3% (в/об) сахарозы, 0,5 мг/л 2,4-D и 7 г бактоагара, pH 5,7. Все эксперименты проводили, начиная с 7 дней после субкультивирования, на основе методики, описанной ниже.
Выделение протопластов
Тридцать миллилитров суспензионной культуры сои сорта Maverick через 7 дней субкультивирования переносили в коническую центрифужную пробирку объемом 50 мл и центрифугировали при 200 g в течение 3 минут с получением приблизительно 10 мл объема осажденных клеток (SCV) в пробирке. Супернатант удаляли, стараясь не затронуть осадок клеток. Двадцать миллилитров раствора ферментов (0,3% пектолиазы (320952; MP Biomedicals), 3% целлюлазы ("Onozuka" R10TM; Yakult Pharmaceuticals, Japan) в растворе MMG (4 мМ MES, 0,6 М маннита, 15 мМ MgCl2, pH 6,0) добавляли на каждые 4 SCV суспензионных клеток и пробирки оборачивали пленкой ParafilmTM. Пробирки устанавливали в шейкер на ночь (приблизительно на 16-18 ч). На следующее утро аликвоту обработанных ферментами клеток тщательно исследовали под микроскопом, чтобы гарантировать, что расщепление клеточных стенок являлось достаточным.
Очистка протопластов
Растворы клеток/ферментов медленно фильтровали через клеточный фильтр с отверстиями 100 мкм. Клеточный фильтр промывали 10 мл среды W5+ (1,82 мМ MES, 192 мМ NaCl, 154 мМ CaCl2, 4,7 мМ KCl, pH 6,0). Этап фильтрования повторяли с использованием фильтра с отверстиями 70 мкм. Конечный объем доводили до 40 мл путем добавления 10 мл среды W5+. Клетки перемешивали переворачиванием пробирок. Протопласты медленно наслаивали на слой 8 мл раствора сахарозы (500 мМ сахарозы, 1 мМ CaCl2, 5 мМ MES-KOH, pH 6,0), добавляя раствор сахарозы на дно конической центрифужной пробирки объемом 50 мл, содержащей клетки. Пробирки центрифугировали при 350×g в течение 15 минут в бакет-роторе. Наконечник на 5 мл для пипетки использовали, чтобы медленно отбирать слой протопластов (приблизительно 7-8 мл). Затем протопласты переносили в коническую пробирку объемом 50 мл и добавляли 25 мл промывочного раствора W5+. Пробирки медленно переворачивали и центрифугировали в течение 10 минут при 200 g. Супернатант удаляли, добавляли 10 мл раствора MMG и медленно переворачивали пробирки, чтобы ресуспендировать протопласты. Плотность протопластов определяли с помощью гемоцитометра или проточного цитометра. Как правило, из 4 PCV суспензии клеток получали приблизительно 2 миллиона протопластов.
Трансформация протопластов с использованием ПЭГ
Концентрацию протопластов доводили до 1,6 миллиона/мл, используя MMG. Аликвоты протопластов по 300 мкл (приблизительно 500000 протопластов) переносили в стерильные пробирки объемом 2 мл. Суспензию протопластов регулярно перемешивали при переносе протопластов в пробирки. Плазмидную ДНК добавляли к аликвотам протопластов согласно схеме эксперимента. Штатив, содержащий пробирки с протопластами, медленно переворачивали 3 раза по 1 минуте для смешивания ДНК и протопластов. Протопласты инкубировали в течение 5 минут при комнатной температуре. Триста микролитров раствора полиэтиленгликоля (ПЭГ 4000) (40% этиленгликоля (81240-Sigma Aldrich), 0,3 М маннита, 0,4 М CaCl2) добавляли к протопластам и штатив с пробирками перемешивали в течение 1 мин и инкубировали в течение 5 мин, с мягким переворачиванием два раза во время инкубирования. Один миллилитр W5+ медленно добавляли в пробирки и штатив с пробирками переворачивали 15-20 раз. Затем пробирки центрифугировали при 350 g в течение 5 мин и удаляли супернатант, не затрагивая осадок. Один миллилитр среды WI (4 мМ MES, 0,6 М маннита, 20 мМ KCl, pH 6,0) добавляли в каждую пробирку и штатив с пробирками мягко переворачивали, чтобы ресуспендировать осадки. Штатив накрывали алюминиевой фольгой и клали набок для инкубирования в течение ночи при 23°С.
Измерение частоты трансформации и сбор протопластов
Определение количества протопластов и эффективность трансформации измеряли при использовании проточного цитометра Quanta Flow CytometerTM (Beckman-Coulter Inc). Приблизительно через 16-18 часов после трансформации, 100 мкл из каждого повтора отбирали, помещали в 96-луночный планшет и разводили 1:1 раствором WI. Повторы ресуспендировали 3 раза и 100 мкл использовали для количественного анализа с помощью проточной цитометрии. Перед передачей образцов на анализ образцы центрифугировали при 200 g в течение 5 мин, супернатанты удаляли и образцы быстро замораживали в жидком азоте. Затем образцы помещали на -80°C в морозильную камеру до обработки для молекулярного анализа.
Трансформация ZFN и донора
Для каждого из выбранных геномных локусов в Таблице 5, протопласты сои трансфицировали конструкциями, содержащими зеленый флуоресцентный белок (gfp) для контроля экспрессии гена, только ZFN, только донор и смесь ZFN и донорной ДНК в соотношении 1:10 (по весу). Общее количество ДНК для трансфекции 0,5 миллиона протопластов составило 80 мкг. Все обработки проводили в трех повторах. Используемый gfp контроль экспрессии гена представлял собой pDAB7221 (Фигура 14, SEQ ID NO:7569), содержащий кассеты экспрессии гена, включающие промотор вируса мозаики жилок маниока-кодирующую последовательность зеленого флуоресцентного белка-3’UTR ORF24 Agrobacterium tumefaciens. Для получения достаточного количества суммарной ДНК на трансфекцию, в качестве наполнителя использовали либо сперму лосося, либо плазмиду, содержащую ген gfp, при необходимости. В стандартном эксперименте таргетинга трансфицировали 4 мкг ZFN, одной или с 36 мкг донорных плазмид, и добавляли нужное количество спермы лосося или ДНК плазмиды pUC19 для доведения общего количества ДНК до конечного количества 80 мкг. Включение плазмиды экспрессии гена gfp в качестве наполнителя позволяет оценить качество трансфекции во множестве локусов и воспроизвести обработки.
Пример 5: Расщепление геномных локусов в сое с помощью цинк-пальцевой нуклеазы
Таргетинг в отобранные геномные локусы демонстрировали посредством вызванного ZFN расщепления ДНК и донорной вставки с применением Системы быстрого таргетинга на основе протопластов (RTA). Для каждого отобранного локуса сои создавали до шести конструкций ZFN и трансформировали ими протопласты, отдельно или с универсальным донорным полинуклеотидом, при этом опосредованное ZFN расщепление и вставку оценивали с использованием Секвенирования следующего поколения (NGS) или соединительной (внутренней-внешней) ПЦР, соответственно.
Трансфицированные ZFN протопласты сои собирали через 24 часа после трансфекции центрифугированием при 1600 об/мин в пробирках EPPENDORFTM объемом 2 мл и полностью удаляли супернатант. Геномную ДНК выделяли из осадка протопластов при использовании набора Qiagen plant DNA extraction kitTM (Qiagen, Valencia, CA). Выделенную ДНК ресуспендировали 50 мкл воды и определяли концентрацию с помощью NANODROP® (Invitrogen, Grand Island, NY). Целостность ДНК оценивали при разгоне образцов на электрофорезе в 0,8% агарозном геле. Все образцы были нормализованы (20-25 нг/мкл) для ПЦР амплификации с целью получения ампликонов для секвенирования (Illumina, Inc., San Diego, CA). ПЦР-праймеры со штрихкодом для амплификации областей, включающих каждую тестируемую последовательность распознавания ZFN из обработанных и контрольных образцов, подбирали и приобретали в IDT (Coralville, IA, очищенные с помощью ВЭЖХ). Оптимальные условия амплификации определяли с помощью градиентной ПЦР при использовании 0,2 мкМ подходящих штрихкод-праймеров ACCUPRIME PFX SUPERMIXTM (Invitrogen, Carlsbad, CA) и 100 нг геномной ДНК-матрицы в реакции объемом 23,5 мкл. Параметры циклов являлись следующими: начальная денатурация при 95°C (5 мин) с последующими 35 циклами денатурации (95°C, 15 сек), отжигом (55-72°C, 30 сек), элонгацией (68°C, 1 мин) и конечной достройкой (68°C, 7 мин). Продукты амплификации анализировали в 3,5% TAE агарозных гелях и определяли подходящую температуру отжига для каждой комбинации праймеров, а затем использовали при амплификации ампликонов из контрольных и обработанных ZFN образцов, как описано выше. Все ампликоны очищали в 3,5% агарозных гелях, элюируемых водой, и концентрации определяли с помощью NANODROPTM. Для секвенирования следующего поколения (Next Generation Sequencing) приблизительно 100 нг ПЦР ампликона из обработанных ZFN и соответствующих необработанных контролей объединяли в пулы и секвенировали с использованием Illumina Next Generation Sequencing (NGS).
Исследовали активность расщепления подходящих ZFN в каждом из оптимальных геномных локусов сои. Короткие ампликоны, содержащие сайты расщепления ZFN, амплифицировали с геномной ДНК и подвергали Illumina NGS из обработанных ZFN и контрольных протопластов. Вызванное ZFN расщепление или двухцепочечный разрыв ДНК устраняли посредством механизма клеточной репарации NHEJ со вставкой или делецией нуклеотидов (инсерции-делеции) на участке расщепления, при этом наличие инсерций-делеций на участке расщепления, таким образом, является показателем активности ZFN, и было определено с помощью NGS. Активность расщепления мишень-специфических ZFN оценивали как количество последовательностей с инсерциями-делециями на 1 миллион последовательностей высокого качества при использовании программы для анализа NGS (патентная публикация 2012-0173,153, анализ данных последовательностей ДНК). Активности наблюдали для выбранных геномных локусов-мишеней в сое и дополнительно подтверждали с помощью выравнивания последовательностей, которые показали разнообразный футпринт инсерций-делеций на каждом сайте расщепления ZFN. Эти данные указывают, что выбранные геномные локусы сои поддаются расщеплению нуклеазами ZFN. Дифференциальная активность на каждой мишени отражает ее состояние хроматина и возможность ее расщепления, а также эффективность экспрессии каждой ZFN.
Пример 6: Быстрый направленный анализ интеграции донорного полинуклеотида
Проверку таргетинга универсальной донорной полинуклеотидной последовательности в выбранные геномные локусы-мишени в сое посредством донорной вставки, опосредованной негомологичным соединением концов (NHEJ), выполняли при использовании полупроизводительного способа Быстрого анализа таргетинга на основе протопластов. Для каждого выбранного геномного локуса-мишени в сое тестировали 3-6 конструкций ZFN, при этом таргетинг оценивали при измерении опосредованного ZFN расщепления с помощью методов секвенирования следующего поколения, а донорную вставку - с помощью соединенительной внутренней-внешней ПЦР (Фиг. 6). Выбранные геномные локусы сои, которые были положительными в обоих анализах, идентифицировали как локусы-мишени.
Быстрый направленный анализ ZFN донорной вставки
С целью определения, могут ли выбранные геномные локусы Zea mays являться мишенью для донорной вставки, конструкцию ZFN и конструкцию универсального донорного полинуклеотида совместно вводили в протопласты сои, которые инкубировали в течение 24 часов перед выделением геномной ДНК для анализа. Если экспрессируемая ZFN была способна разрезать связывающий сайт-мишень и в выбранных геномных локусах-мишенях в сое, и в доноре, линеаризованный донор встраивался в расщепленный сайт-мишень в геноме кукурузы посредством механизма негомологичного соединения концов (NHEJ). Подтверждение направленной интеграции в выбранные геномные локусы-мишени сои выполняли на основе стратегии "внутренней-внешней" ПЦР, где "внутренний" праймер распознает последовательность в нативных оптимальных геномных локусах, а "внешний" праймер связывается с последовательностью в донорной ДНК. Праймеры подбирают таким образом, что только в том случае, когда донорная ДНК встраивается в выбранные геномные локусы-мишени сои, ПЦР анализ будет давать продукт амплификации ожидаемого размера. Анализ внутренней-внешней ПЦР выполняли на 5'- и на 3'-концах соединения вставки. Праймеры, используемые для анализа интегрированных донорных полинуклеотидных последовательностей, представлены в Таблице 9.
ZFN донорная вставка в локусы-мишени с использованием гнездовой "внутренней-внешней" ПЦР
Все ПЦР амплификации проводили с использованием набора TaKaRa Ex Taq HSTM (Clonetech, Mountain View, CA). Первую внутреннюю-внешнюю ПЦР проводили в конечном объеме реакции 20 мкл, которая содержала 1× буфер TaKaRa Ex Taq HSTM, 0,2 мМ дНТФ, 0,2 мкМ "внешнего" праймера, 0,05 мкМ "внутреннего" праймера (подобранного к универсальной донорной кассете, описанной выше), 0,75 единицы полимеразы TaKaRa Ex TAQ HSTM и 6 нг выделенной ДНК протопластов сои. Реакцию затем проводили при использовании программы ПЦР, которая состояла из следующего: 94°C в течение 3 мин, 14 циклов при 98°C в течение 12 сек и при 60°C в течение 30 сек, 72°C в течение 1 мин, затем 72°C в течение 10 мин и выдерживание при 4°C. Полученные в результате продукты ПЦР разгоняли в агарозном геле с маркером 1KB PLUS DNA LADDERTM (Life Technologies, Grand Island, NY) для визуализации.
Гнездовую внутреннюю-внешнюю ПЦР проводили в конечном объеме реакции 20 мкл, которая содержала 1× буфер TaKaRa Ex Taq HSTM, 0,2 мм дНТФ, 0,2 мкМ "внешнего" праймера (Таблица 9), 0,1 мкМ "внутреннего" праймера (подобранного к универсальной донорной кассете, описанной выше, Таблица 10), 0,75 единицы полимеразы TAKARA EX TAQ HSTM и 1 мкл первого продукта ПЦР. Реакцию затем проводили при использовании программы ПЦР, которая состояла из следующего: 94°C в течение 3 мин, 30 циклов при 98°C в течение 12 сек, 60°C в течение 30 сек и 72°C в течение 45 сек, затем 72°C в течение 10 мин и выдерживание при 4°C. Полученные в результате продукты ПЦР разгоняли в агарозном геле с маркером 1KB PLUS DNA LADDERTM (Life Technologies, Grand Island, NY) для визуализации.
Список всех "внешних" праймеров для гнездового внутреннего-внешнего ПЦР анализа оптимальных геномных локусов.
Список всех "внутренних" праймеров для гнездоаого внутреннего-внешнего ПЦР анализа оптимальных геномных локусов.
Применение внутреннего-внешнего ПЦР анализа в системе таргетинга протопластов представляло особую сложность, поскольку для трансфекции использовали большое количество плазмидной ДНК, при этом большое количество ДНК оставалось в системе таргетинга протопластов и впоследствии было выделено вместе с клеточной геномной ДНК. Остаточная плазмидная ДНК может уменьшать относительную концентрацию геномной ДНК и снижать общую чувствительность обнаружения, а также может быть существенной причиной неспецифичных, аберрантных реакций ПЦР. Вызванная ZFN и основанная на NHEJ донорная вставка обычно проходит в прямой или обратной ориентации. Внутренний-внешний ПЦР анализ ДНК в случае прямой ориентации вставки часто демонстрирует ложноположительные полосы, возможно обусловленные общими областями гомологии вокруг связывающего сайта ZFN в мишени и доноре, что может приводить к связыванию с праймером и удлинению неинтегрированной донорной ДНК во время процесса амплификации. Ложноположительные сигналы не наблюдались в анализах, в которых исследовали продукты вставки в обратной ориентации, и поэтому все направленные анализы донорной интеграции проводили с исследованием обратной донорной вставки в RTA. Для дополнительного повышения специфичности и снижения фона также применяли стратегию гнездовой ПЦР. В стратегии гнездовой ПЦР использовали вторую реакцию ПЦР амплификации, в которой амплифицировали более короткую область в первом продукте амплификации первой реакции ПЦР. Использование неравных количеств "внутренних" и "внешних" праймеров оптимизировало соединительную ПЦР еще больше для быстрого анализа таргетинга в выбранных геномных локусах.
Результаты внутреннего-внешнего ПЦР анализа визуализировали в агарозном геле. Для всех выбранных геномных локусов сои в Таблице 12 "обработки ZFN + донор" давали полосу близкого к ожидаемому размера на 5'- и 3'-концах. Контрольные обработки только ZFN или только донором были отрицательными по ПЦР, что указывает на то, что способ давал специфичную оценку донорной интеграции в сайте-мишени по меньшей мере 32 из оптимальных негенных геномных локусов сои. Все обработки проводили в 3-6 повторах, при этом присутствие ожидаемого продукта ПЦР в нескольких повторах (≥2 на обоих концах) использовали для подтверждения таргетинга. Донорная вставка посредством NHEJ часто дает побочные продукты с более низкой интенсивностью, которые образуются в результате процессинга линеаризованных концов в сайтах ZFN мишени и/или донора. Кроме того, наблюдали, что различные ZFN обеспечивали различные уровни эффективности при направленной интеграции, причем некоторые ZFN обеспечивали неизменно высокие уровни донорной интеграции, некоторые ZFN обеспечивали менее стабильные уровни донорной интеграции, и другие ZFN не обеспечивали интеграции. В целом, для каждого из выбранных геномных локусов-мишеней сои, которые были исследованы, направленная интеграция была продемонстрирована в репрезентативных геномных локусах-мишенях сои одной или более ZFN, что подтверждает, что каждый из этих локусов мог служить мишенью. Кроме того, каждый из выбранных геномных локусов-мишеней сои подходил для точной генной трансформации. Проверку этих выбранных геномных локусов-мишеней сои повторяли несколько раз с получением аналогичных результатов каждый раз, подтверждая, таким образом, воспроизводимость процесса проверки, который включает создание плазмиды и конструкции, трансформацию протопластов, обработку образца и анализ образца.
Выводы
Донорную плазмиду и одну из ZFN, предназначенную для специфичного расщепления выбранных геномных локусов-мишеней сои, трансфицировали в протопласты сои и через 24 часа собирали клетки. Анализ геномной ДНК, выделенной из контрольных, обработанных ZFN и обработанных ZFN и донором протопластов, при использовании внутренней-внешней соединительной ПЦР показал наличие направленной вставки универсального донорного полинуклеотида в результате расщепления геномной ДНК под действием ZFN (Таблица 12). Эти исследования показывают, что универсальная донорная полинуклеотидная система может применяться для оценки таргетинга на эндогенных участках и для скрининга кандидатных ZFN. Наконец, быстрый анализ таргетинга на основе протопластов и новые системы универсальных донорных полинуклеотидных последовательностей обеспечивают быстрый путь для скрининга геномных мишеней и ZFN для работ в области точной геномной инженерии в растениях. Способы могут быть дополнены для оценки сайт-специфического расщепления и донорной вставки в геномных мишенях в любой представляющей интерес системе при использовании любой нуклеазы, которая вводит двухцепочечные или одноцепочечные разрывы ДНК.
Более 7018 выбранных геномных локусов были идентифицированы по различным критериям, подробно описанным выше. Выбранные геномные локусы разделили на кластеры при использовании анализа главных компонент, основанного на десяти параметрах, используемых для определения выбранных геномных локусов. Репрезентативные кластеры в дополнение к некоторым другим целевым локусам, как продемонстрировали, могут служить в качестве мишеней.
Представлены результаты интеграции универсальной донорной полинуклеотидной последовательности в выбранные геномные локусы-мишени в сое. Как указано знаком * ниже, донорная вставка в OGL37 была подтверждена только реакцией ПЦР 5’ и 3’ соединительной последовательности.
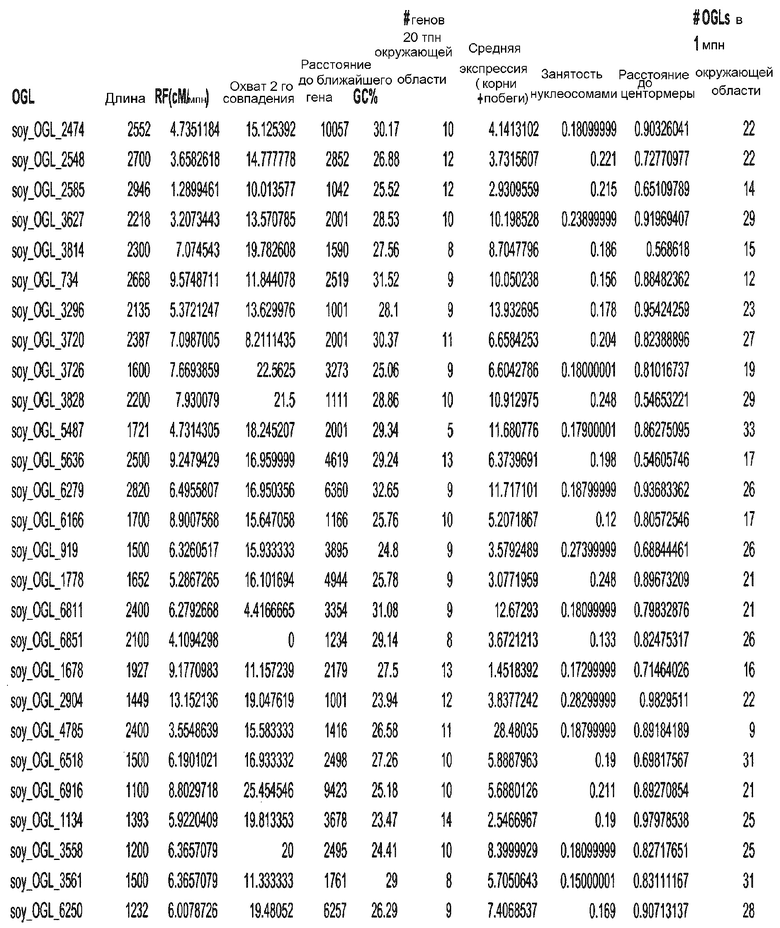
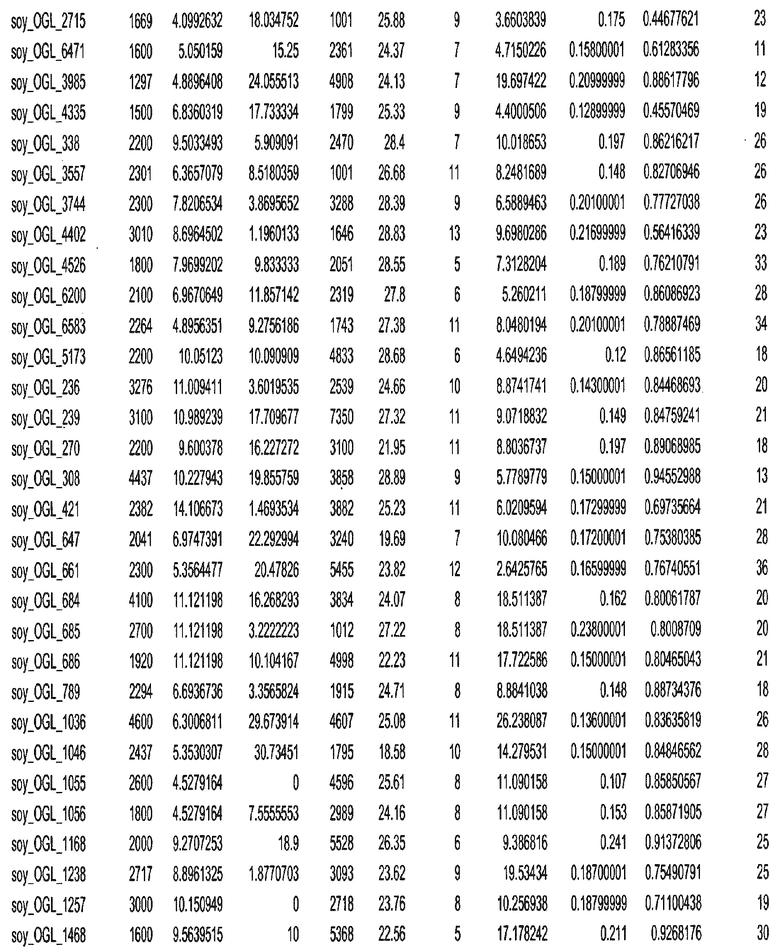

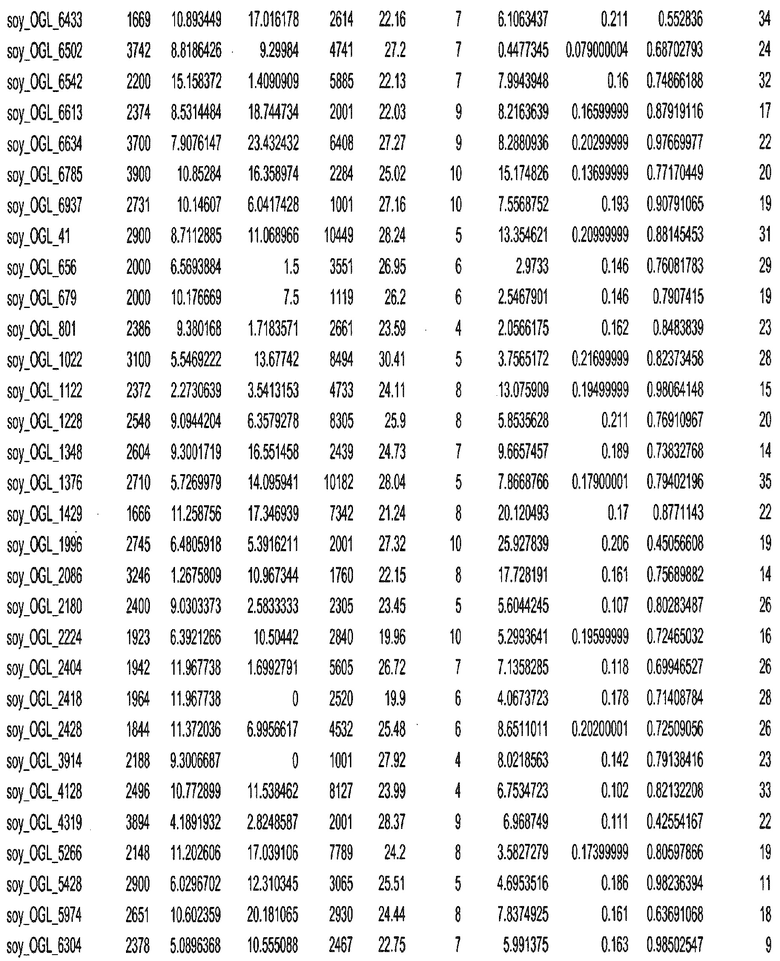
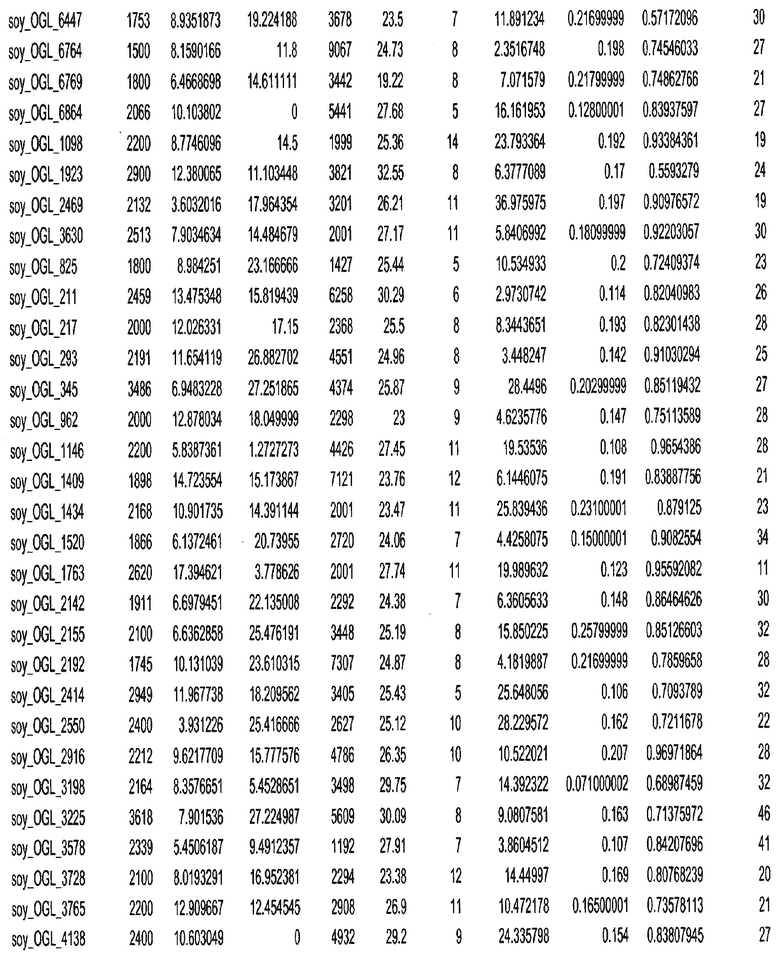
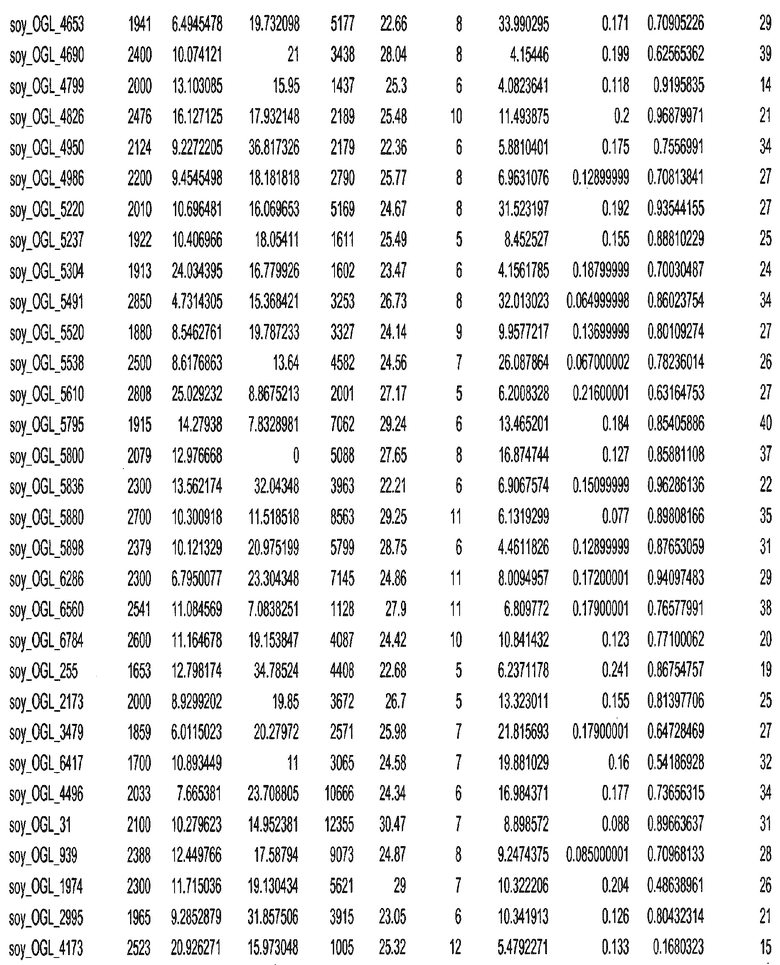
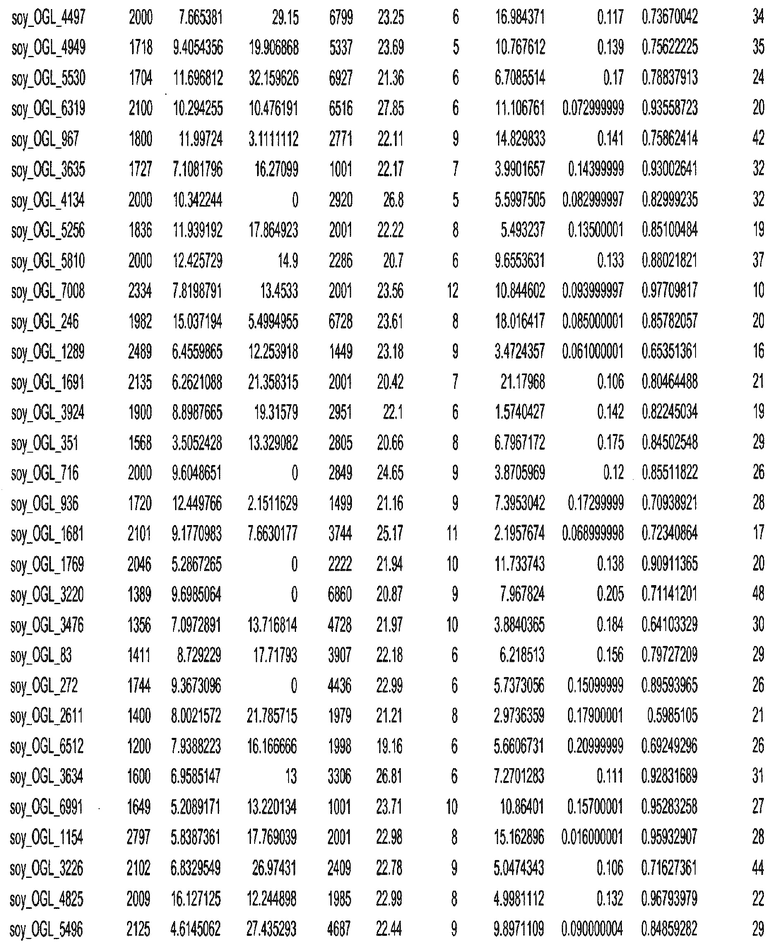

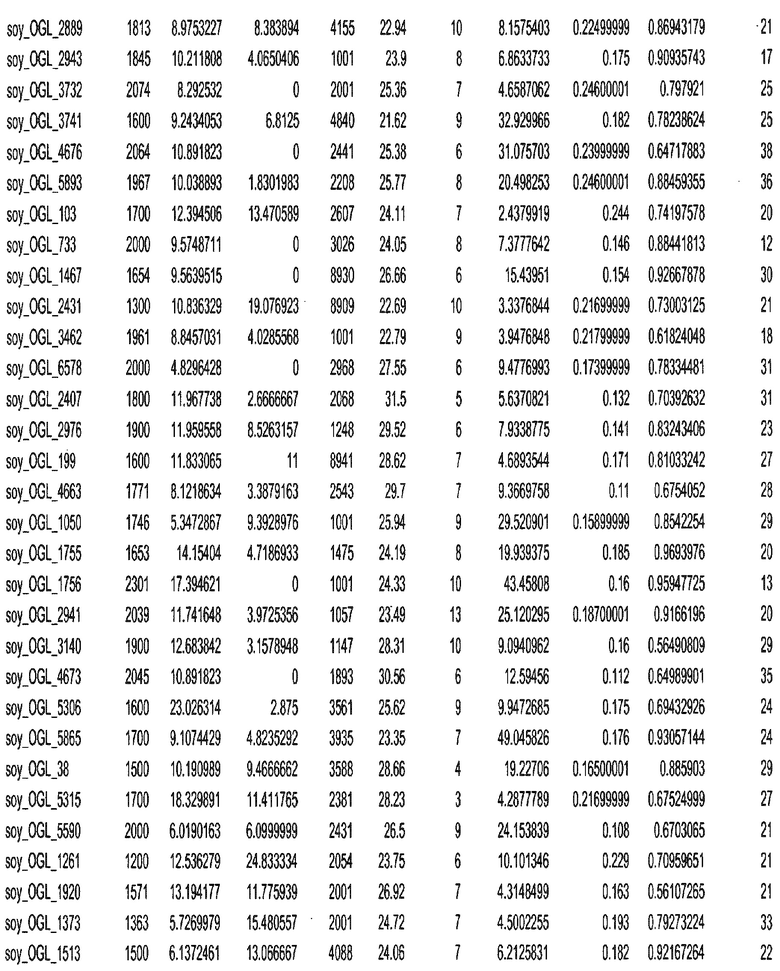
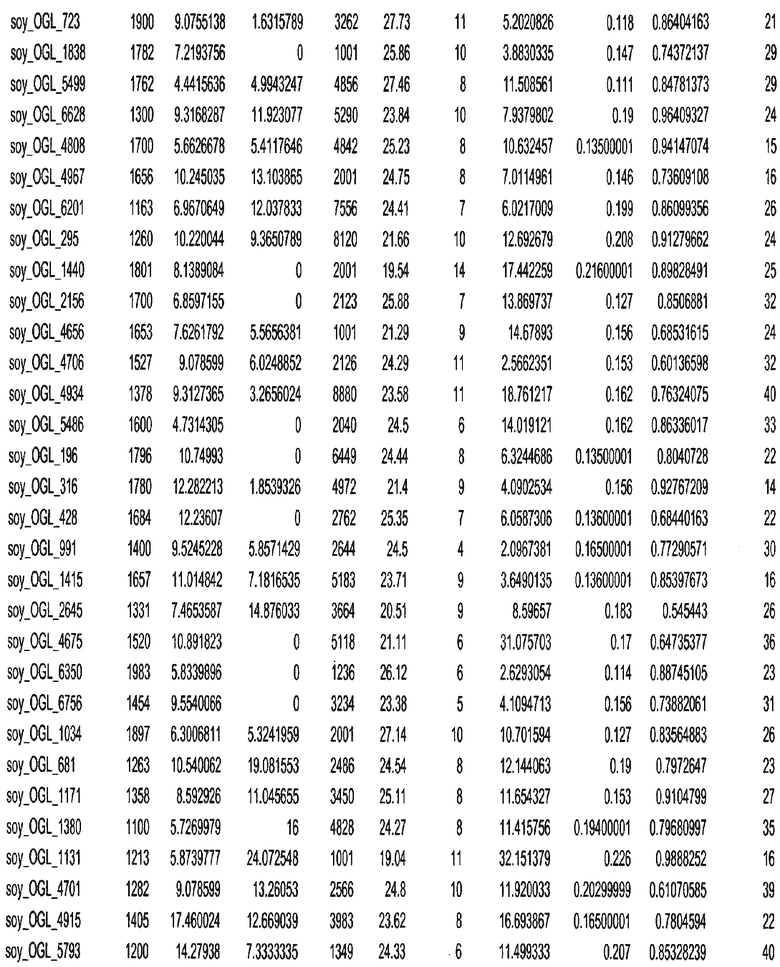
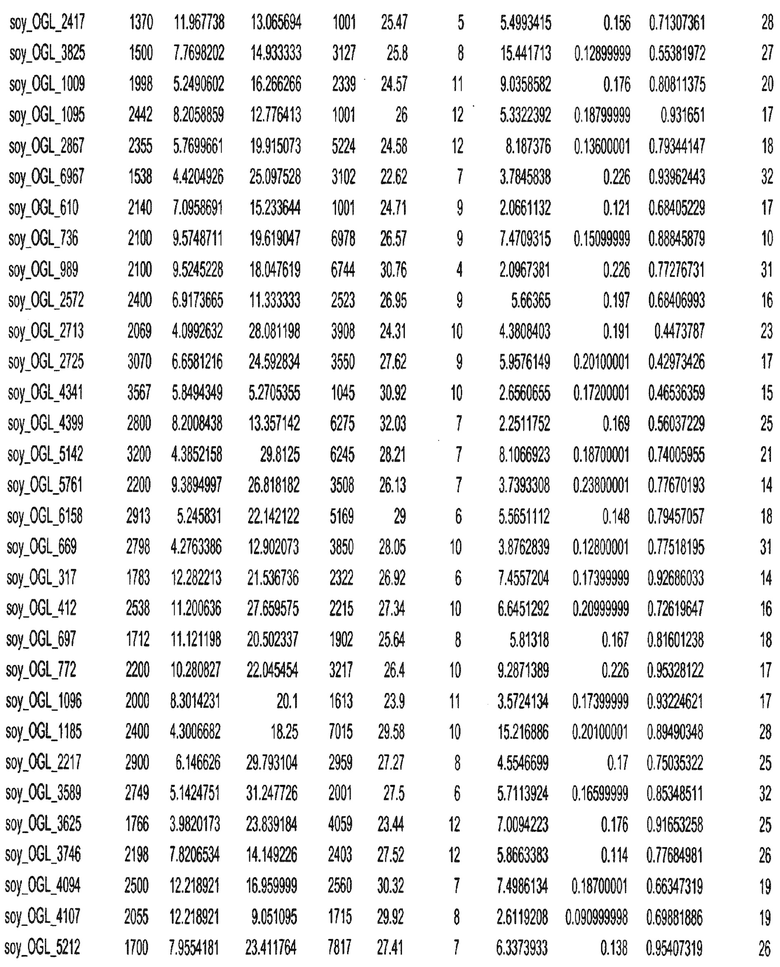
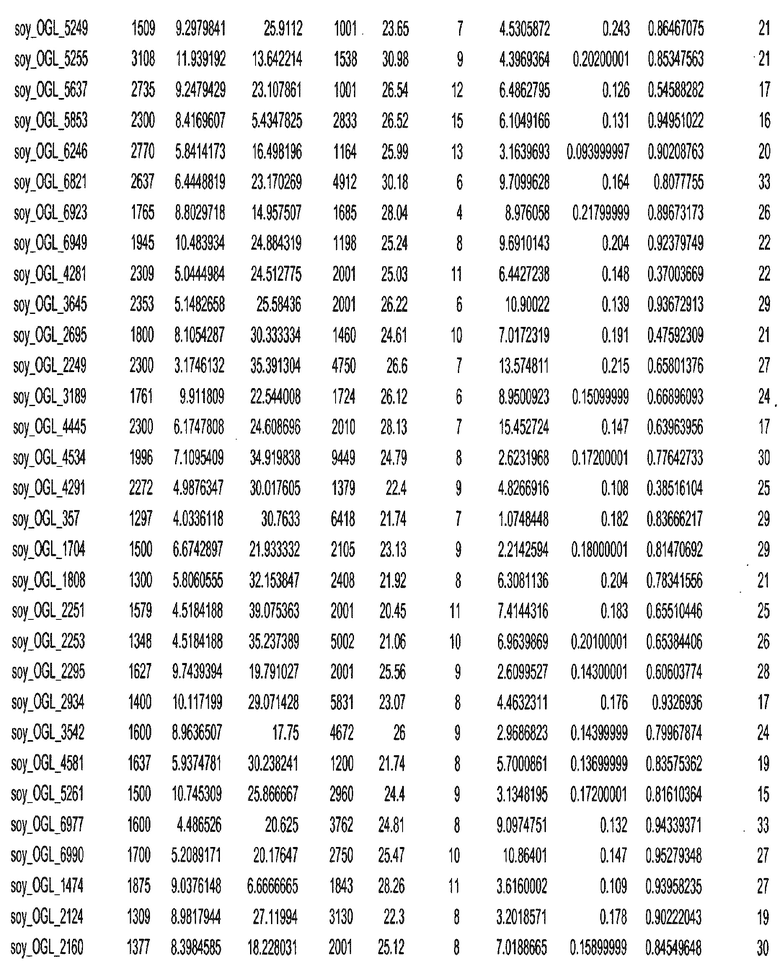
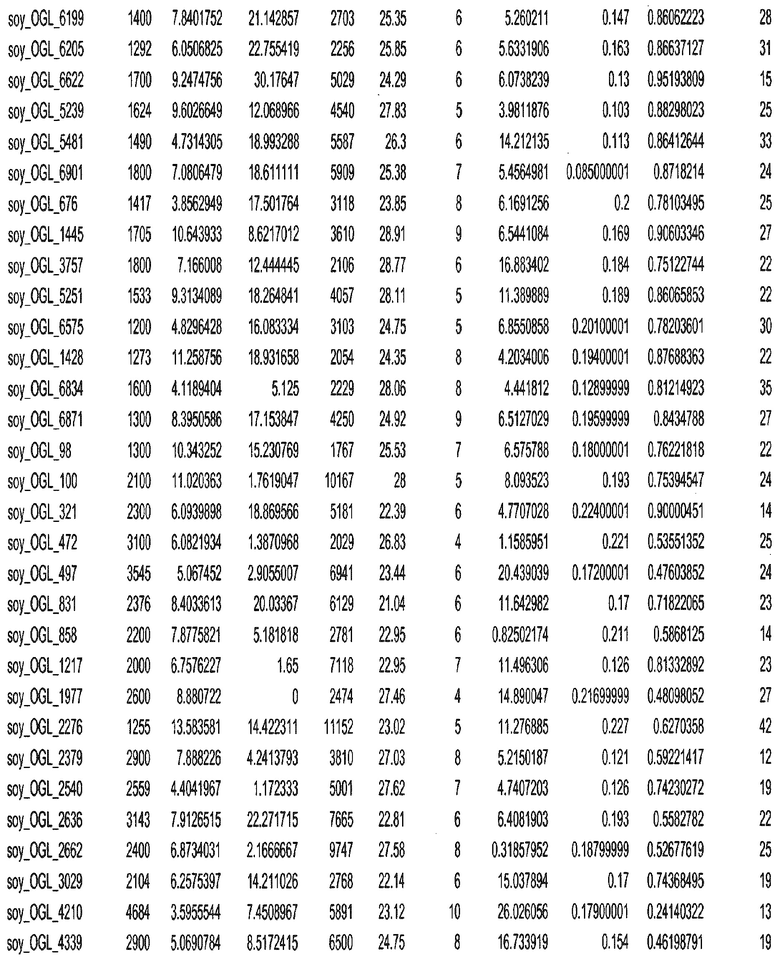
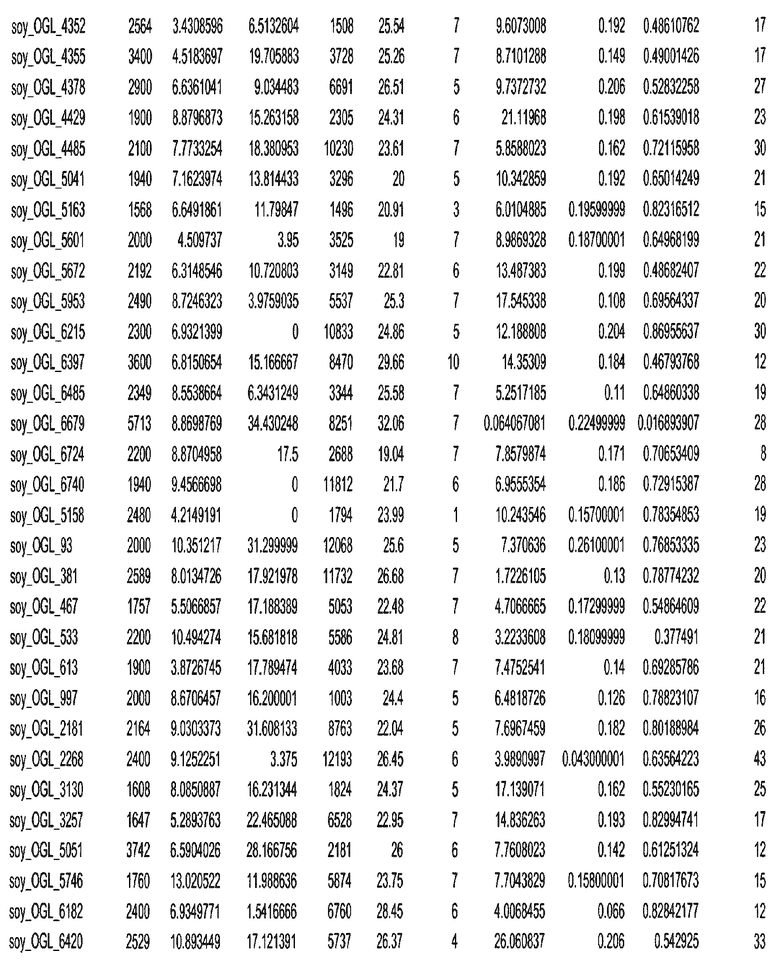
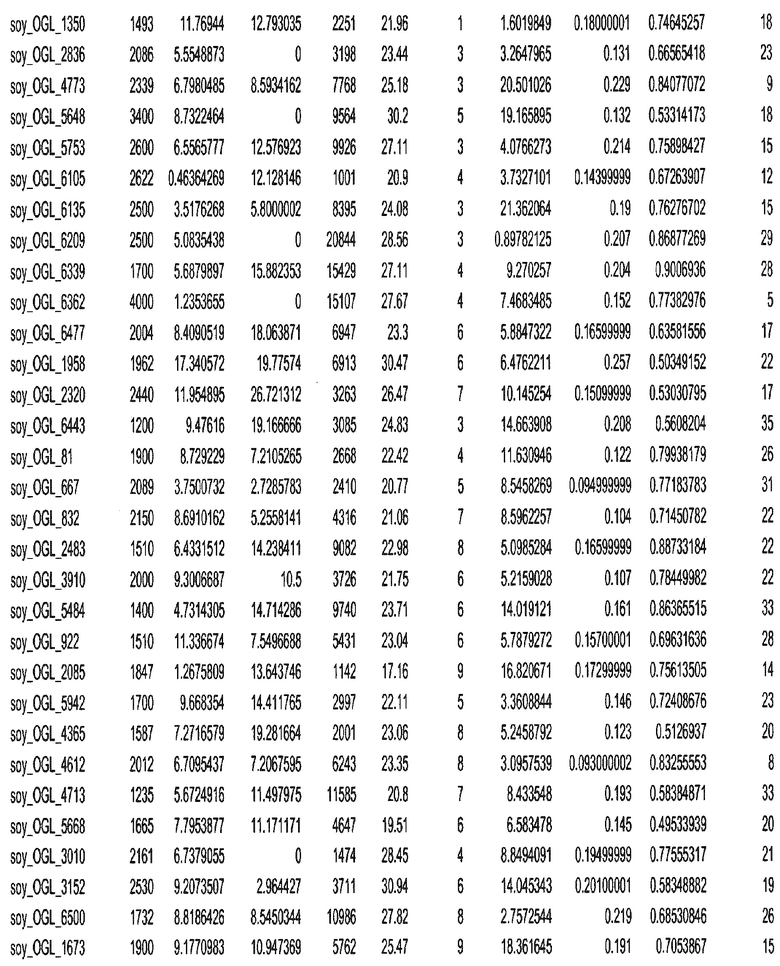
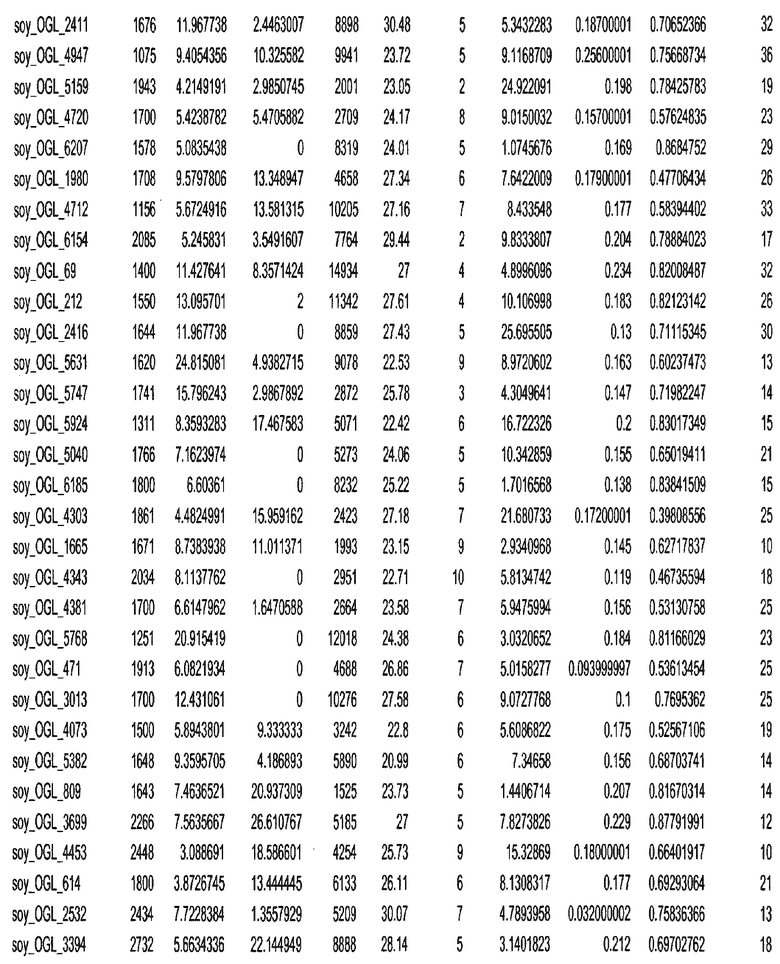
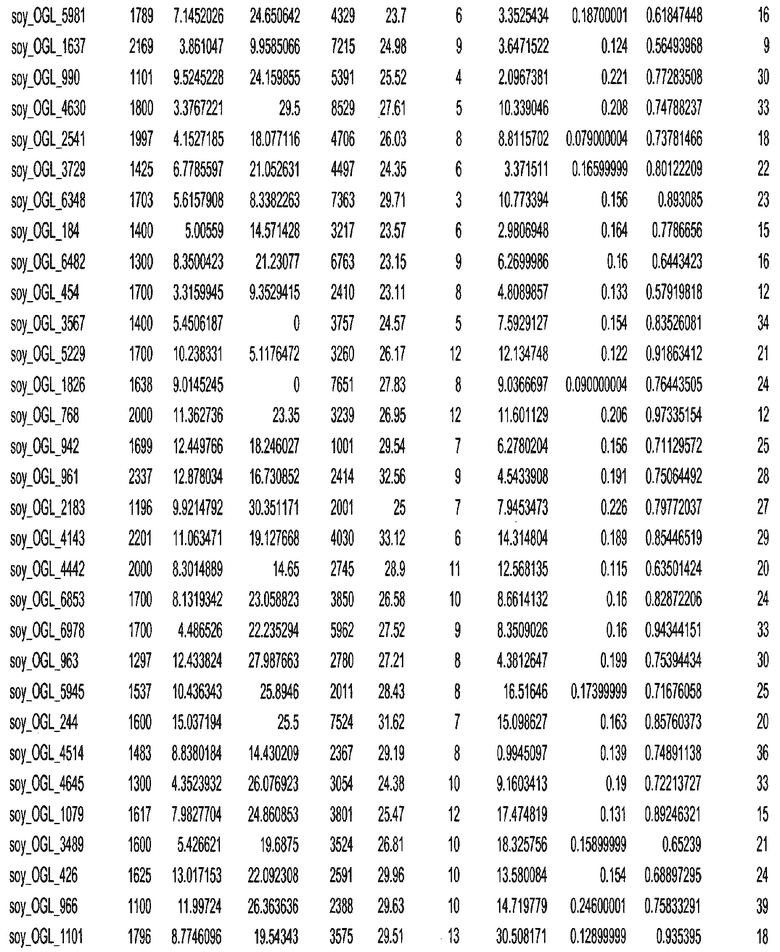
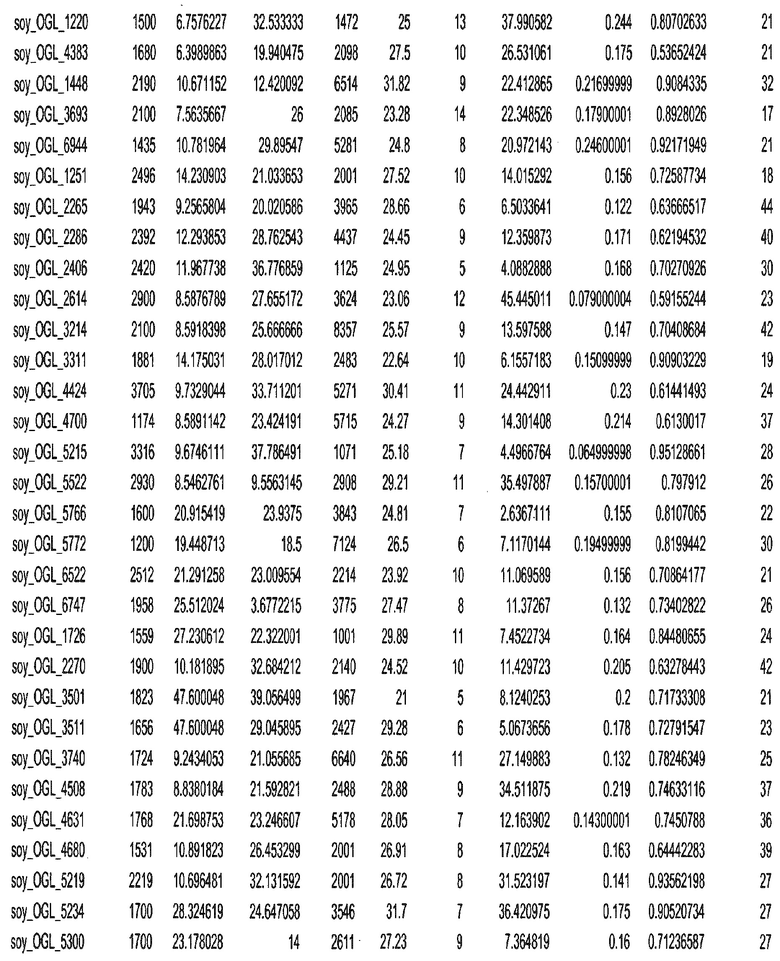
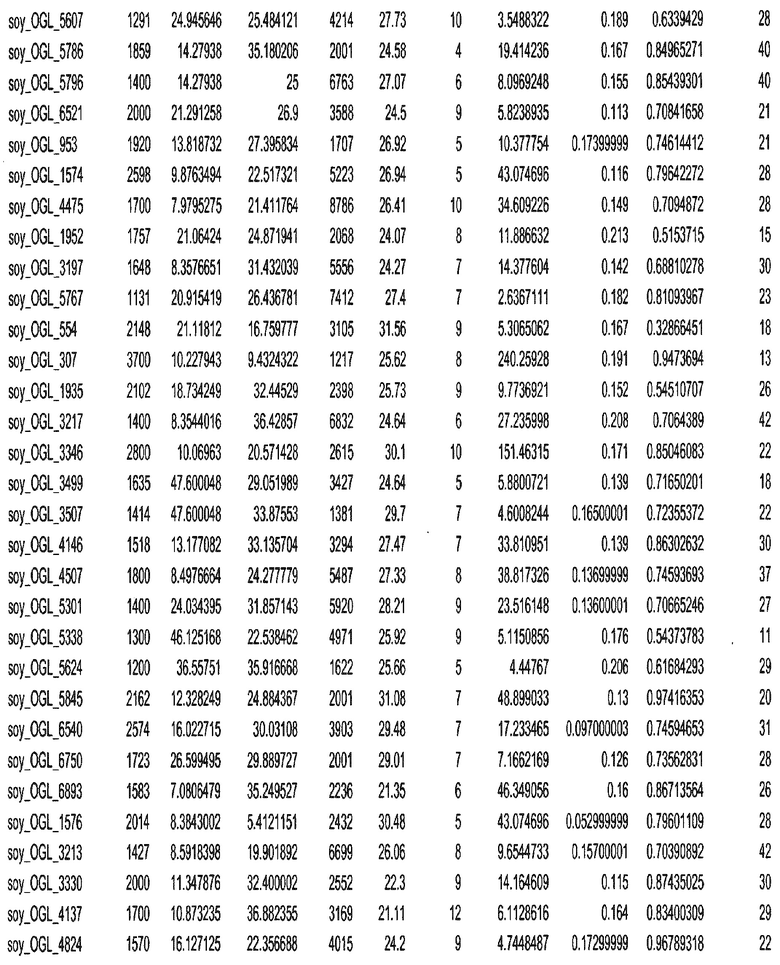

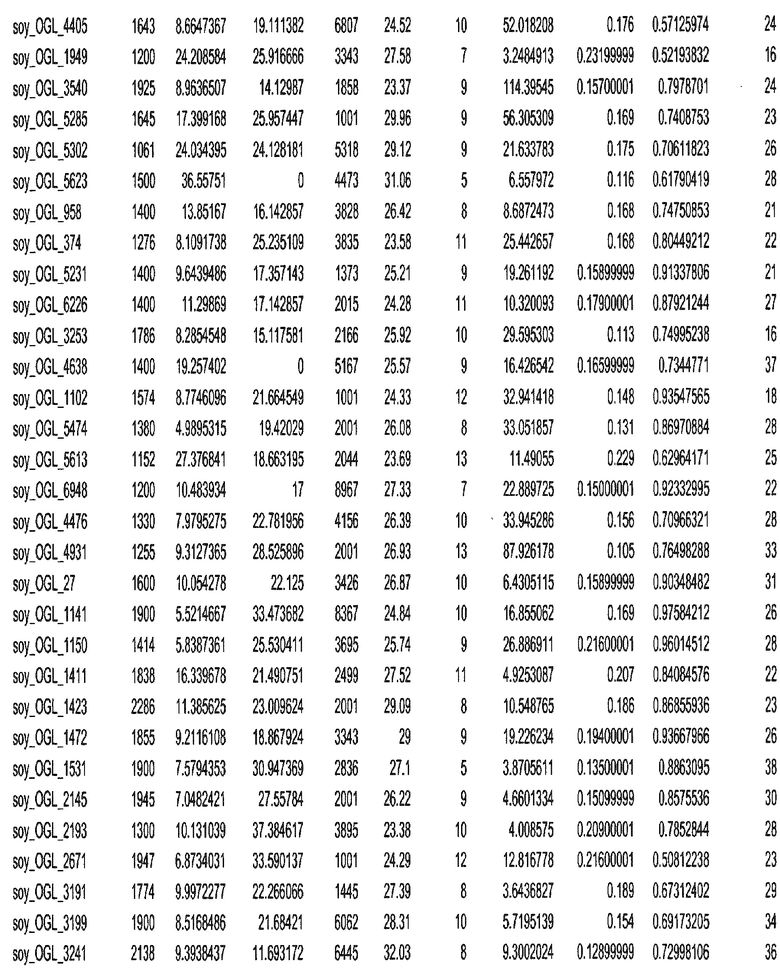
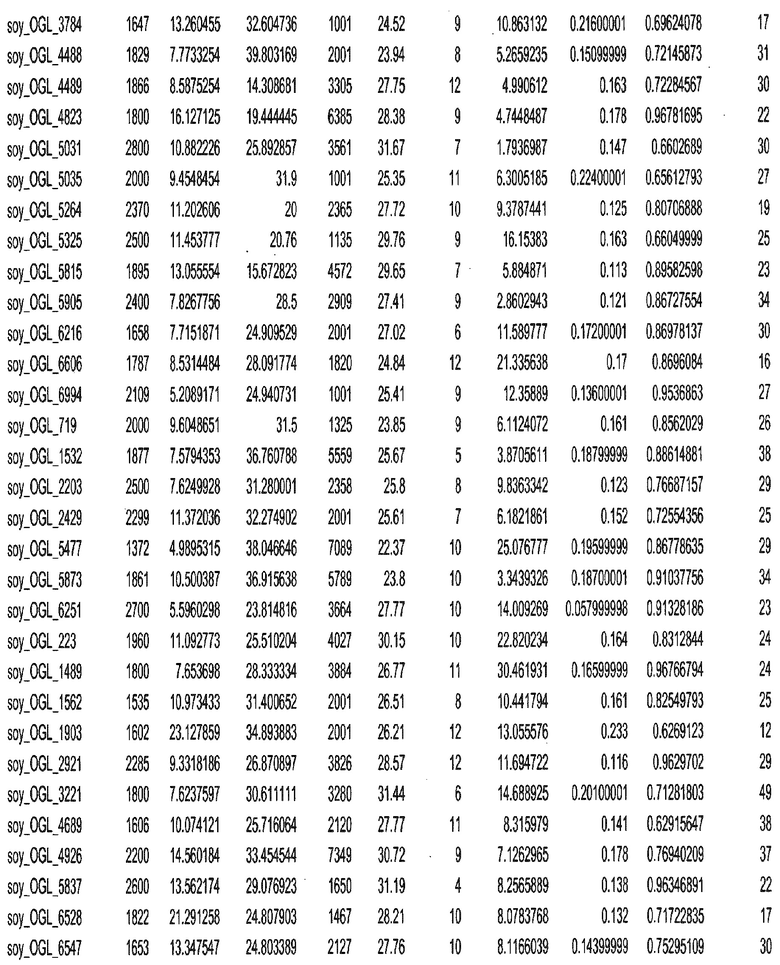
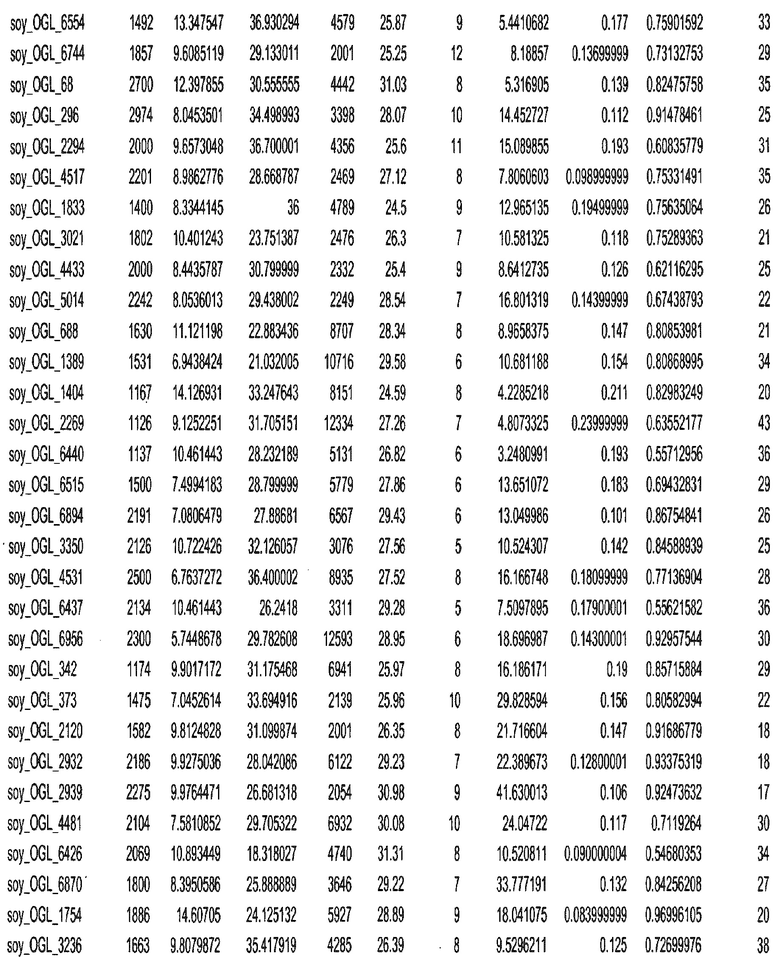
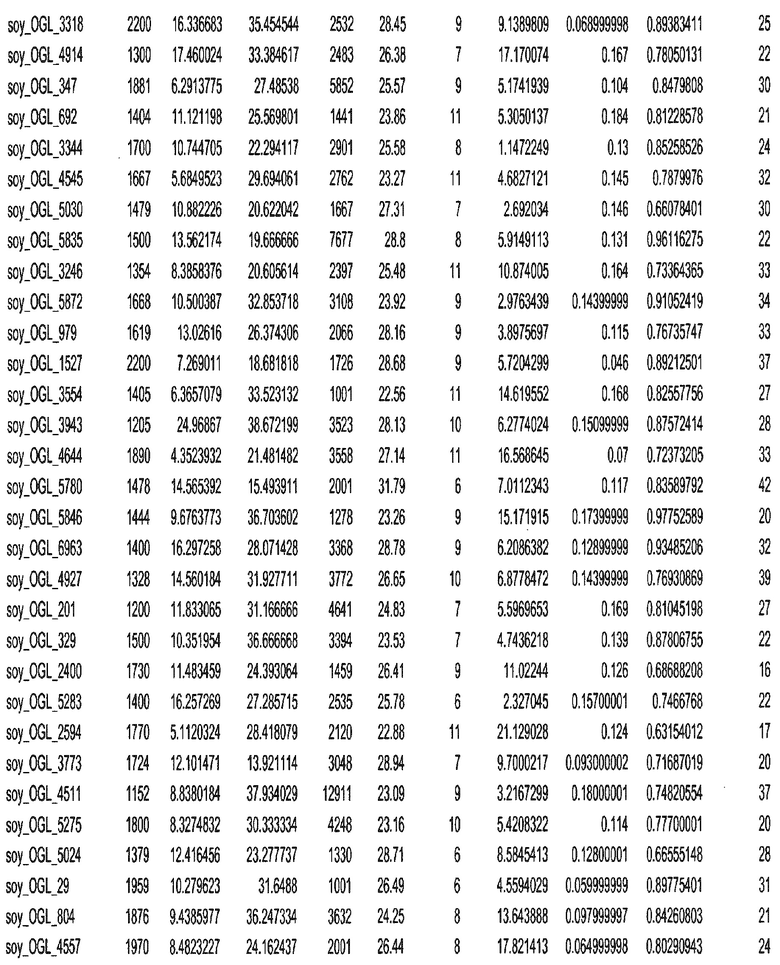
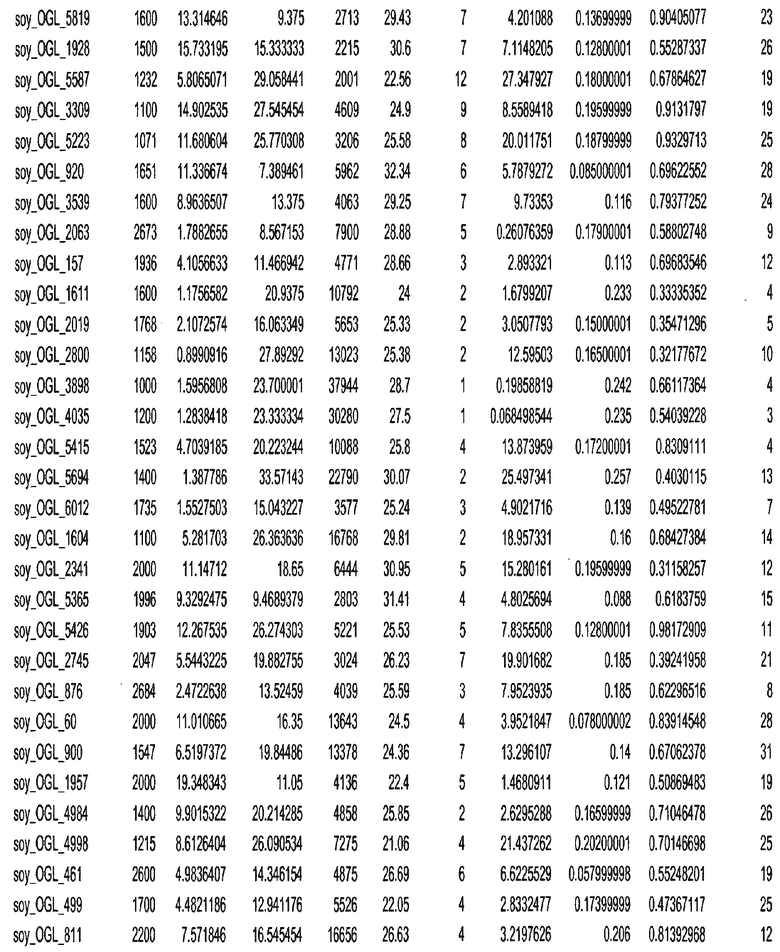
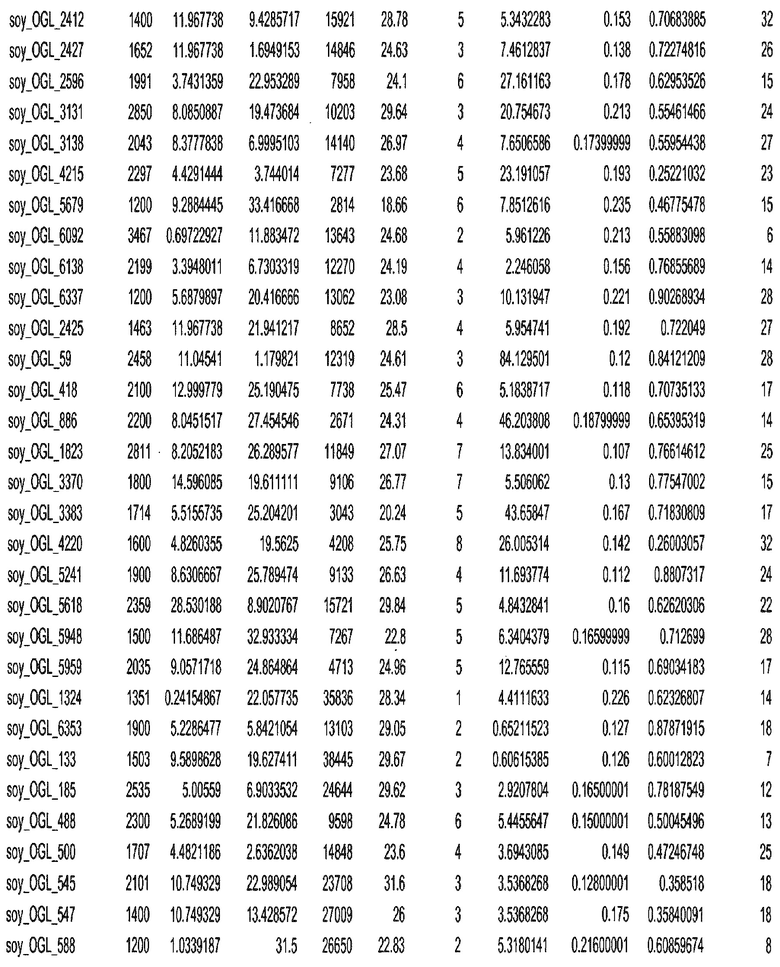
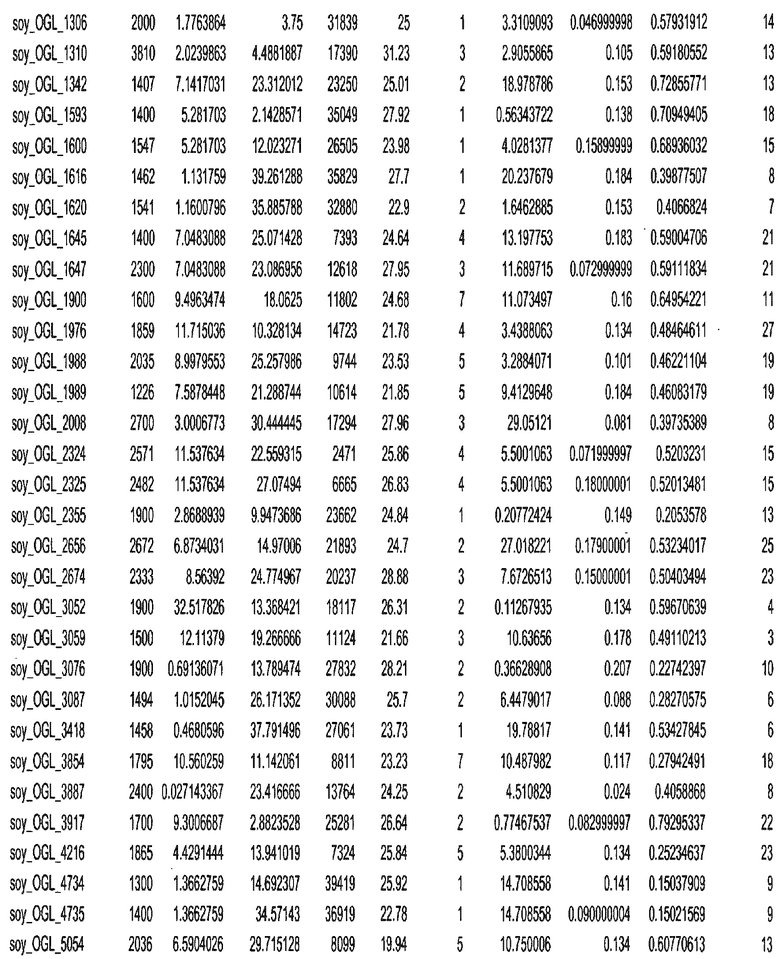
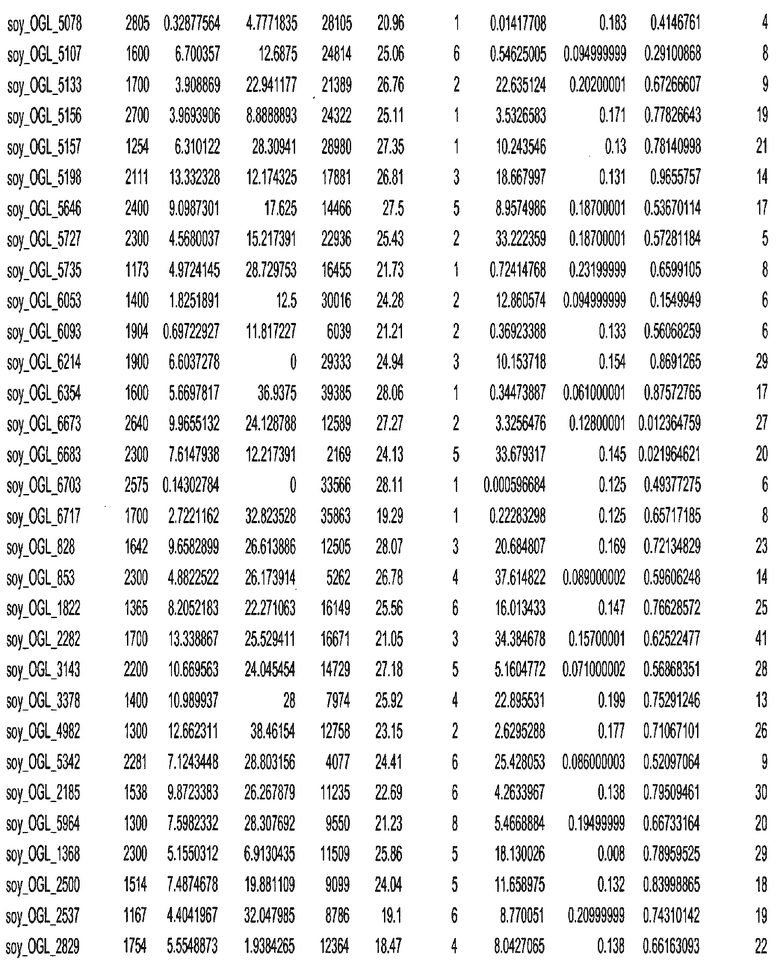
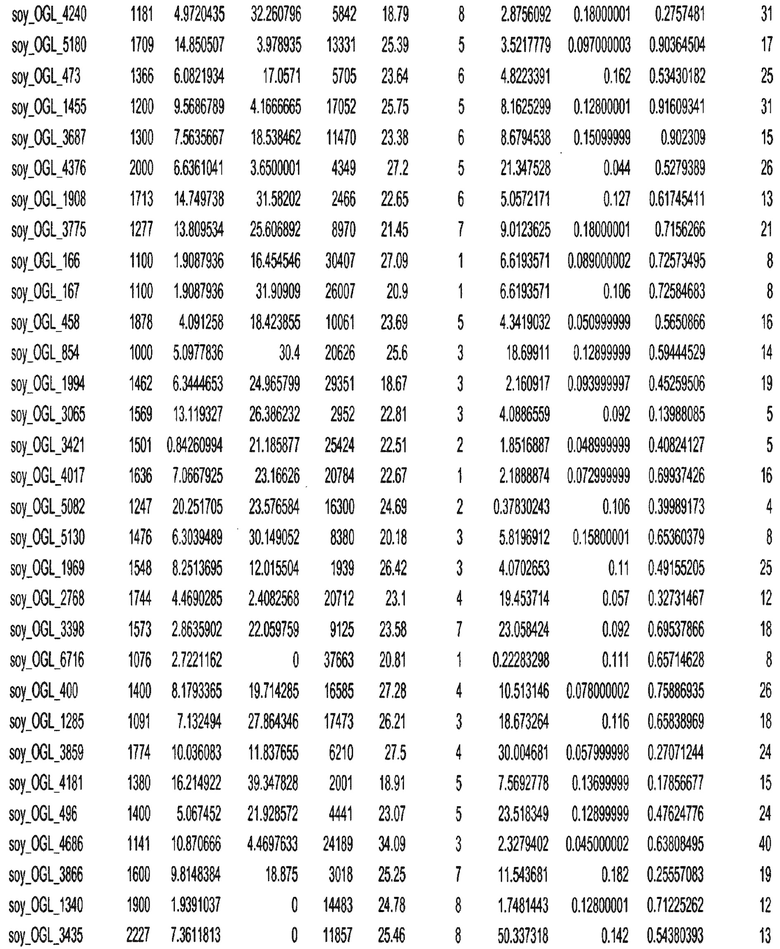
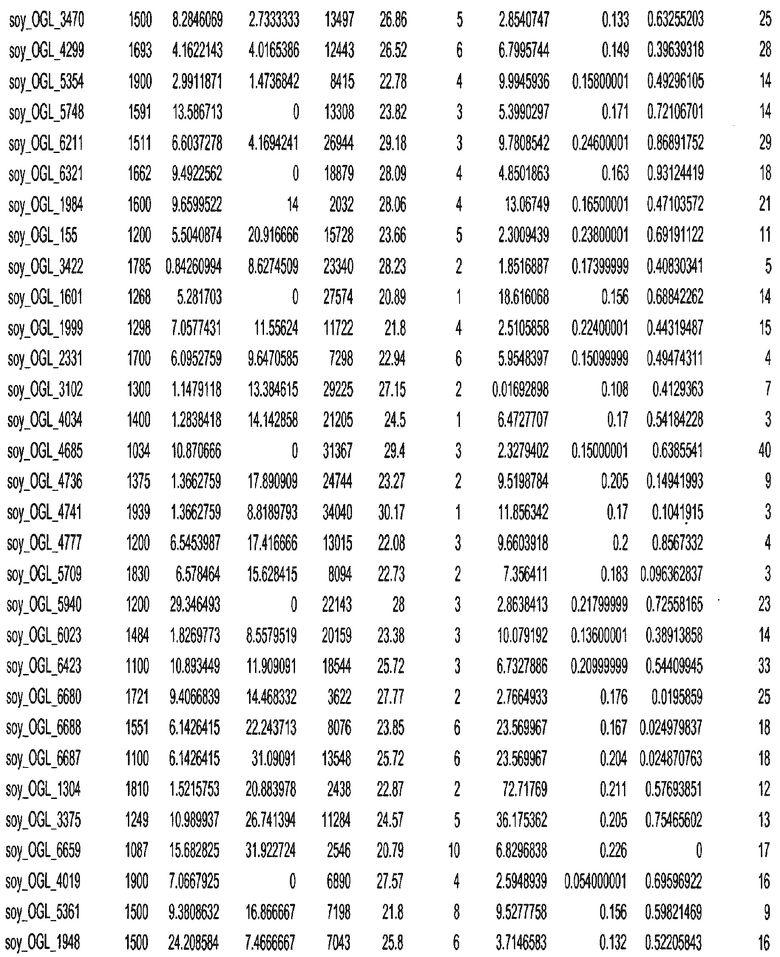
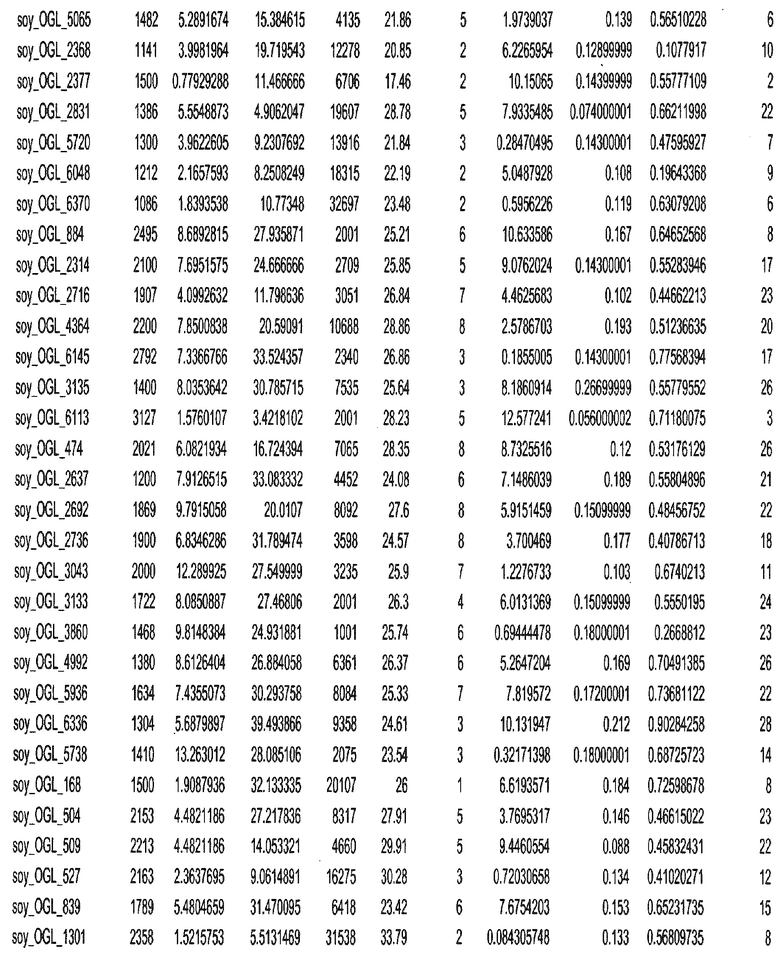
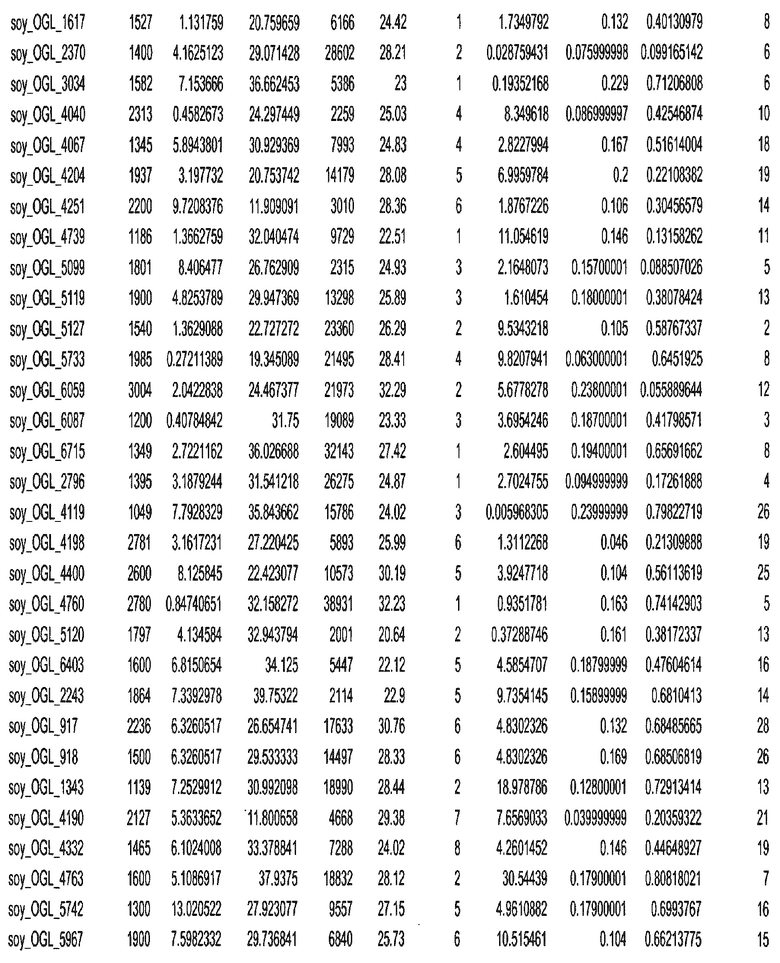
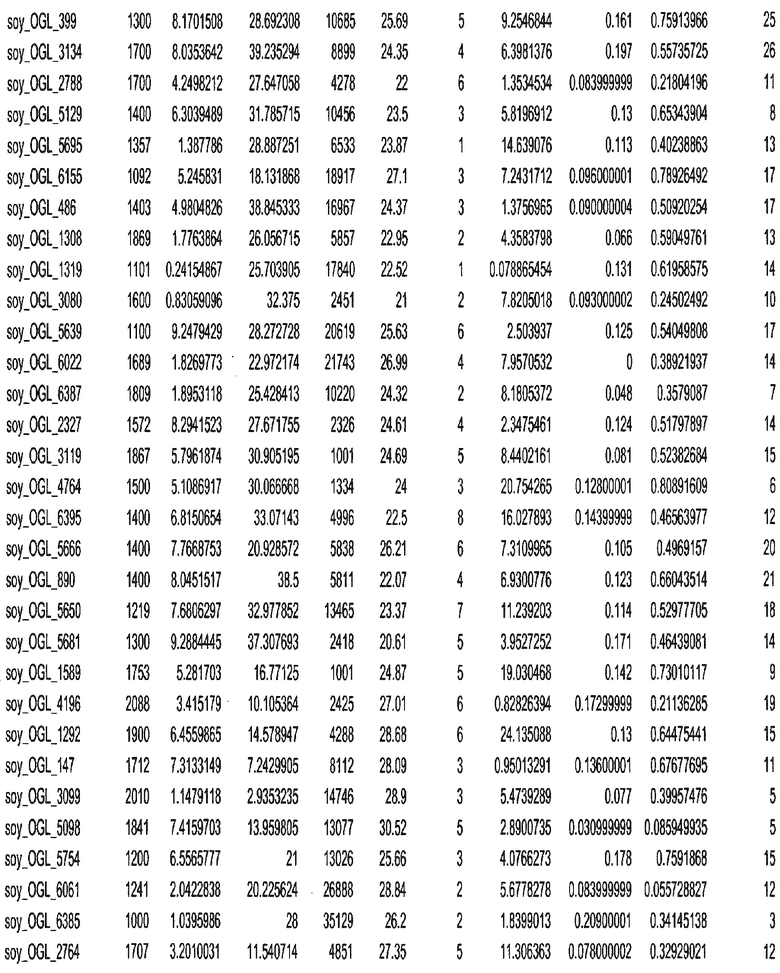
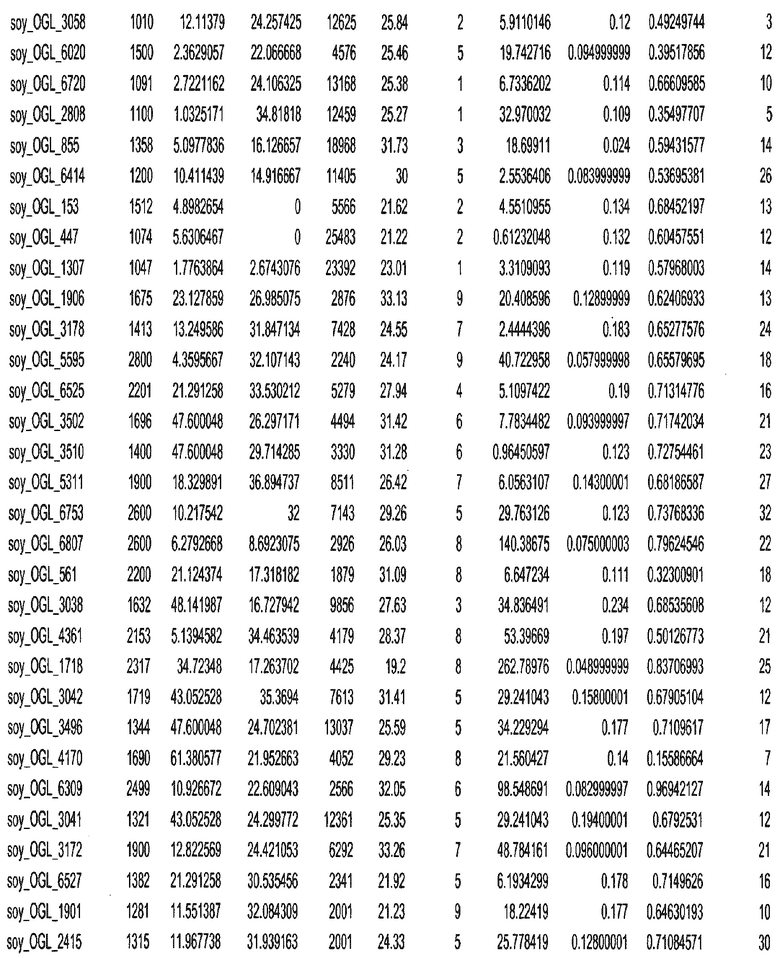

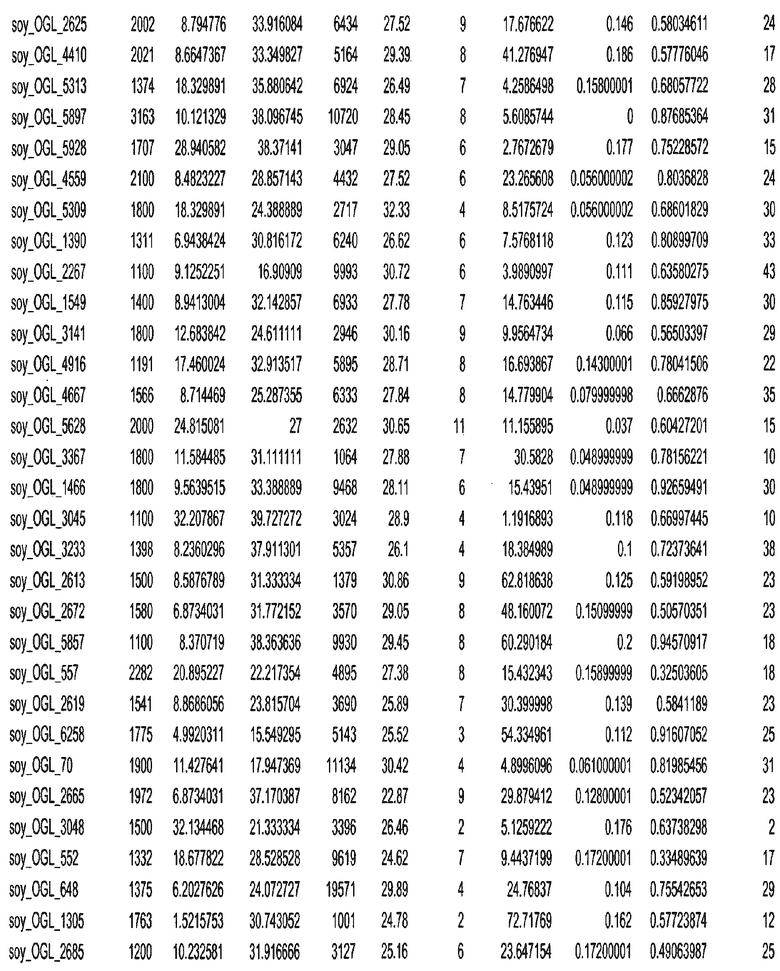
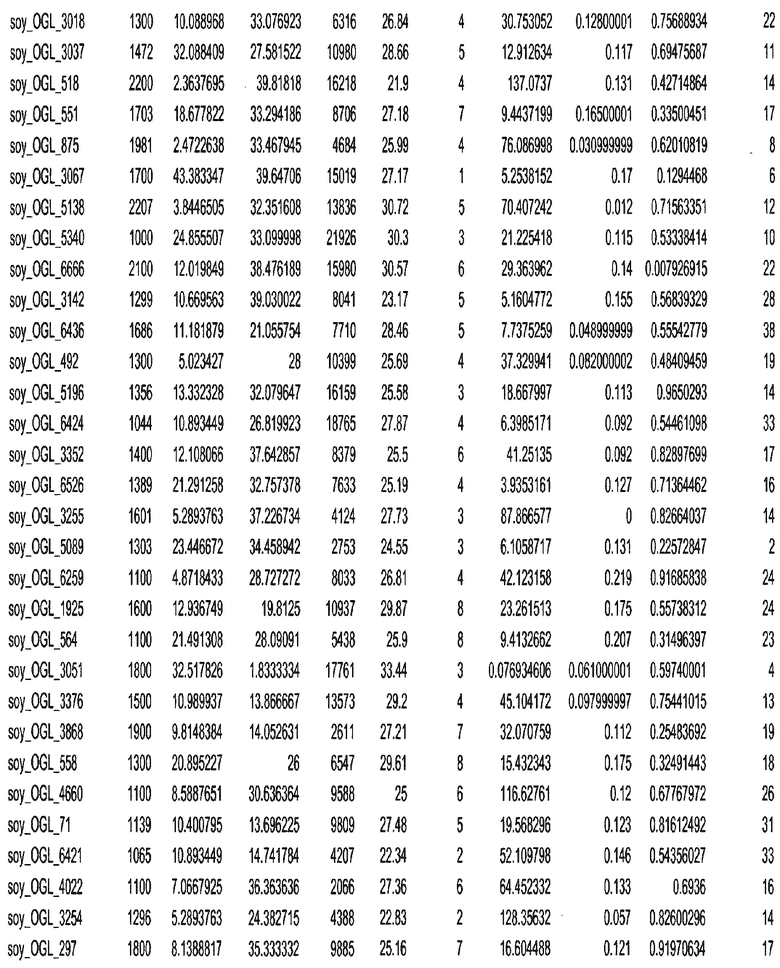
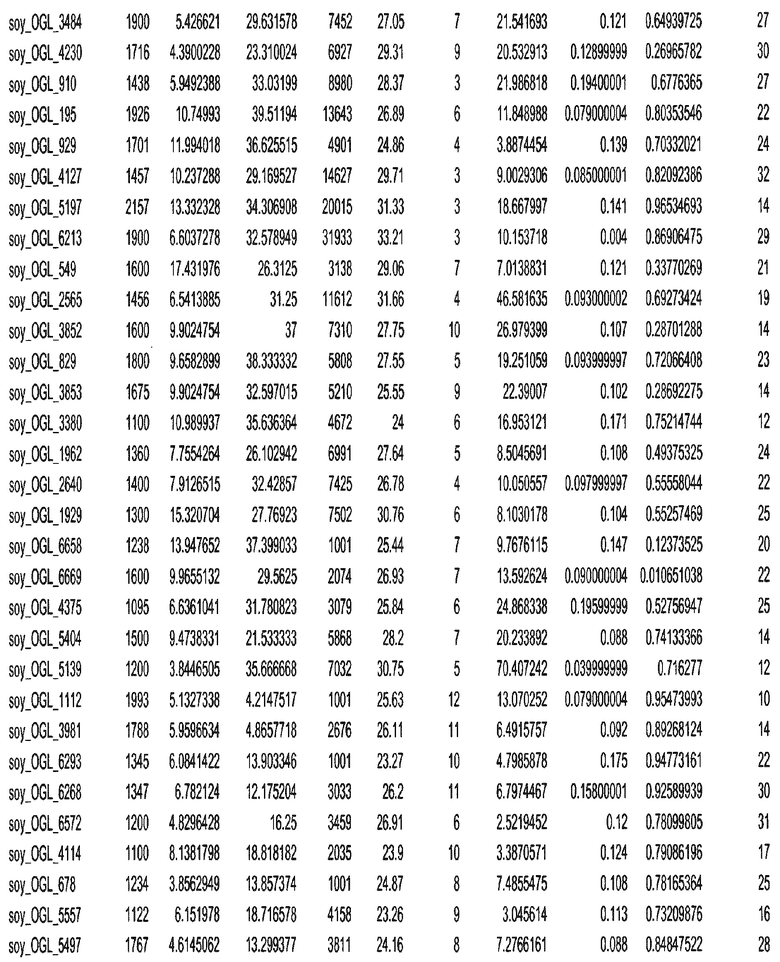
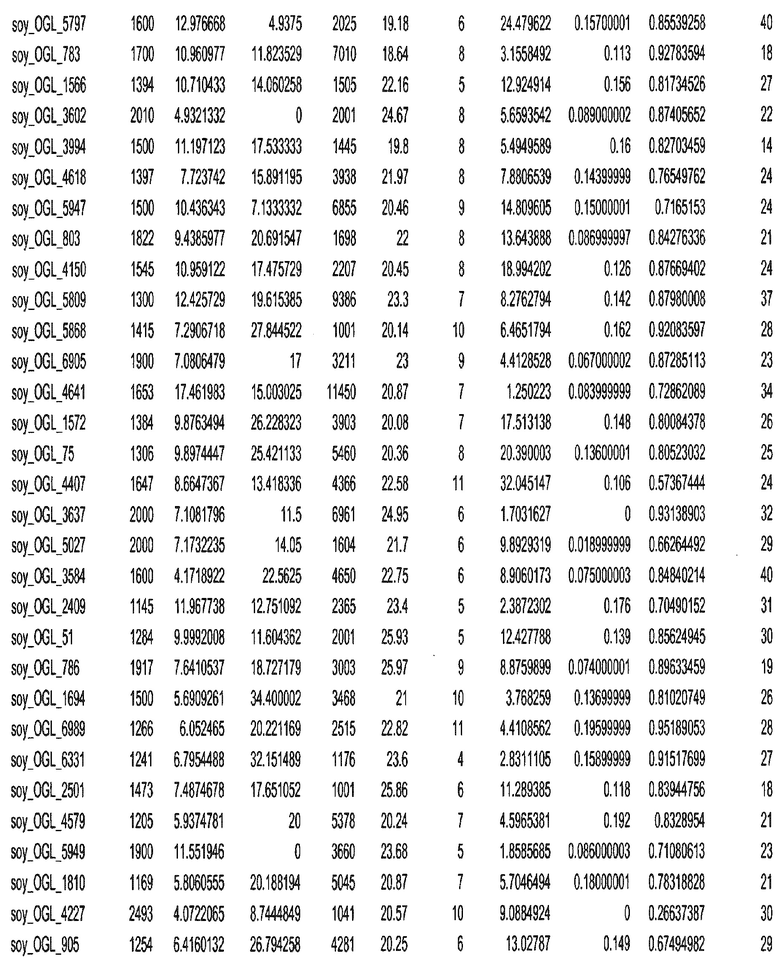
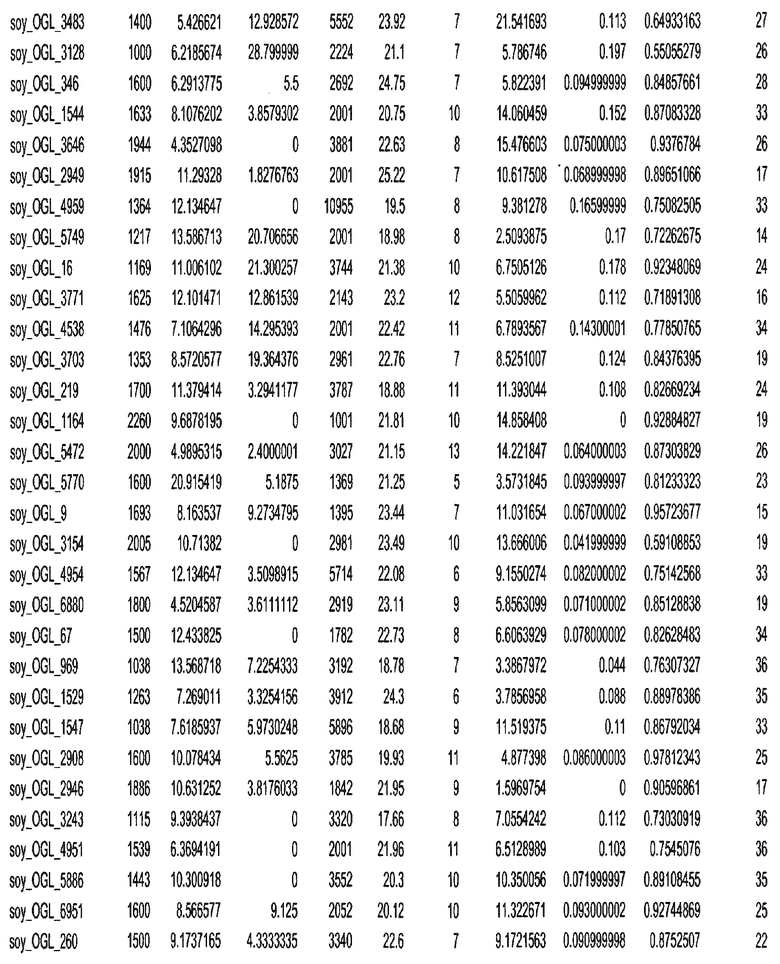
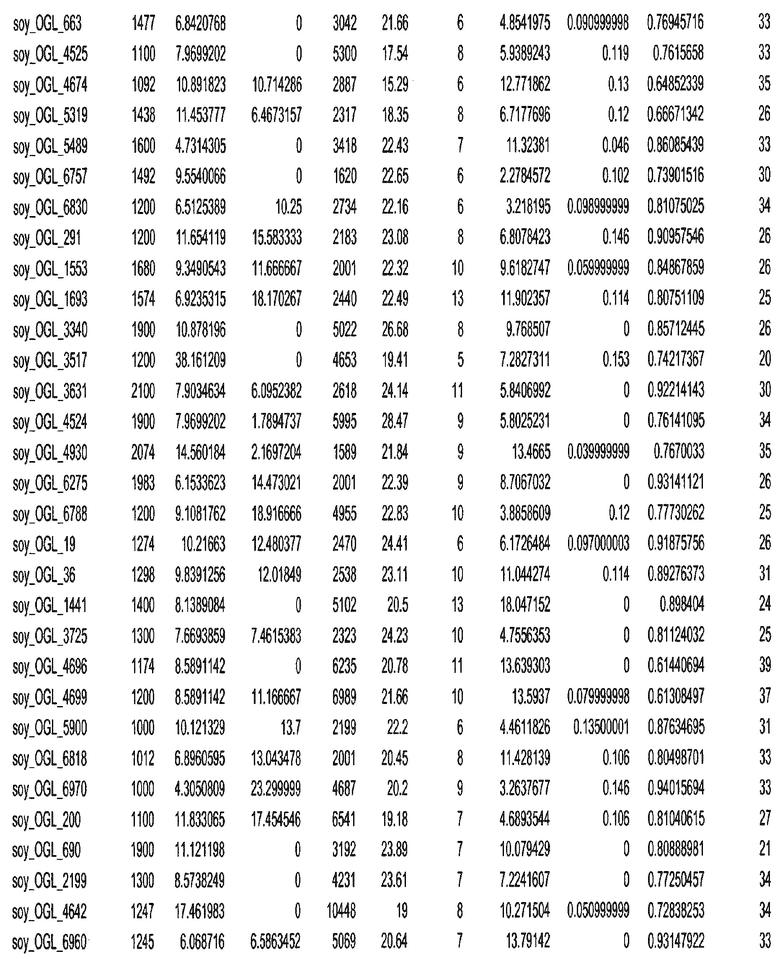
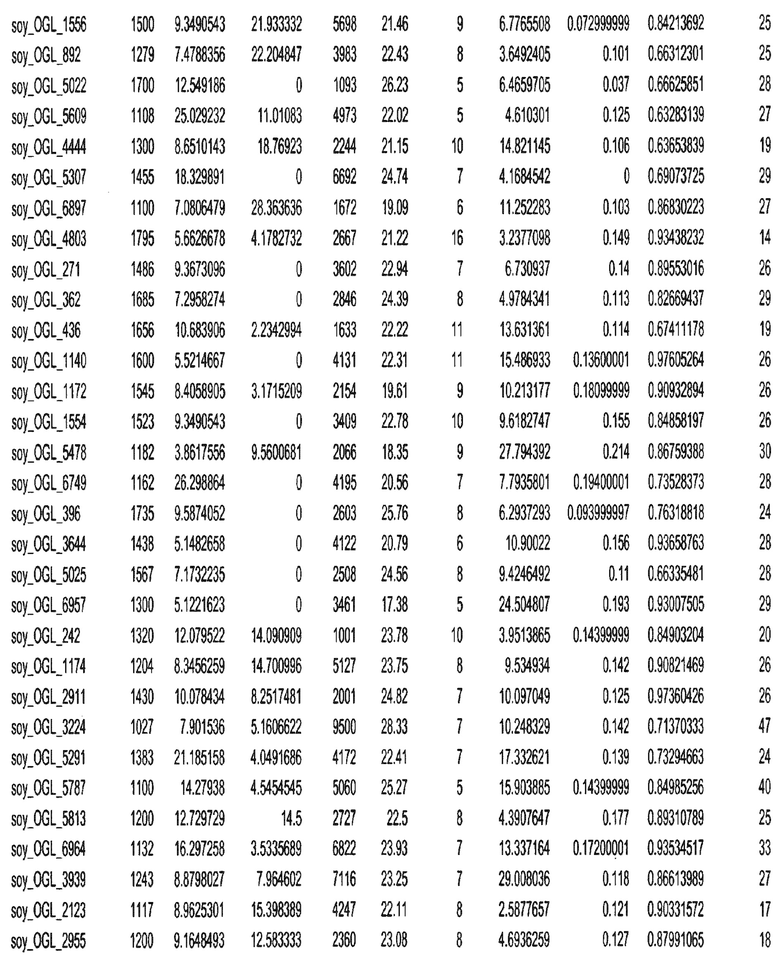

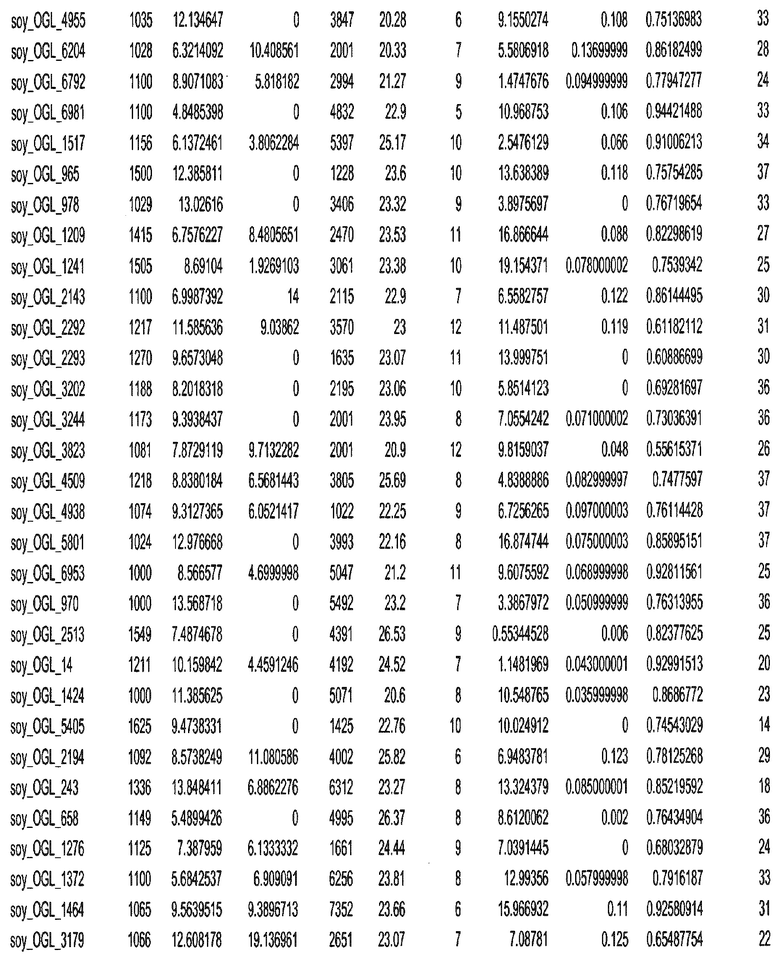
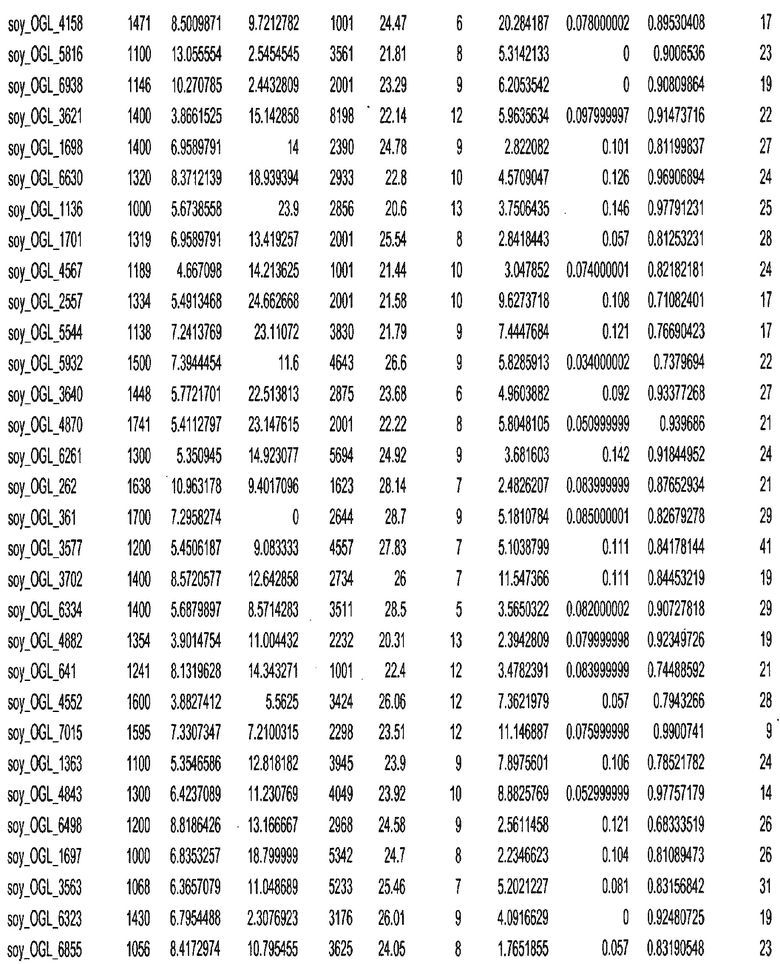
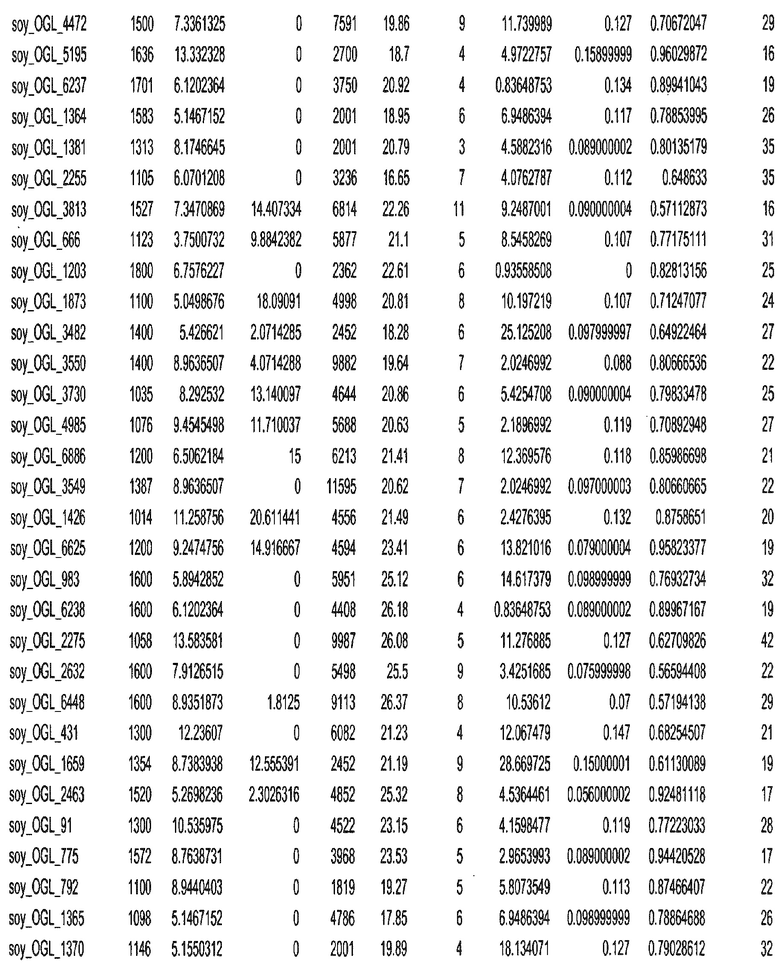
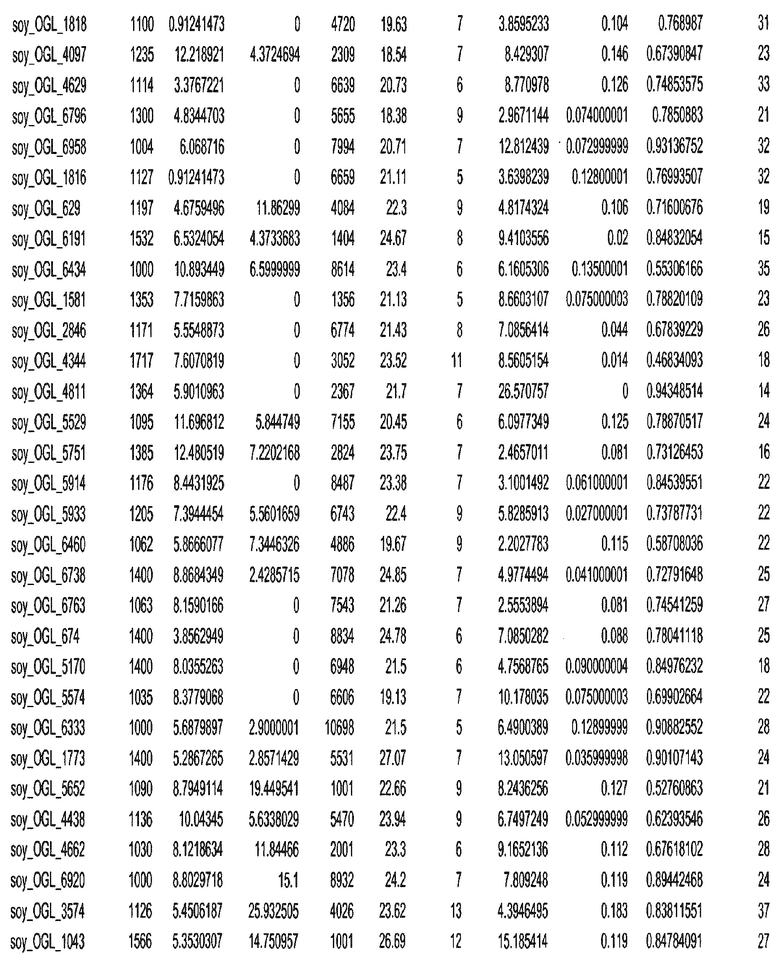
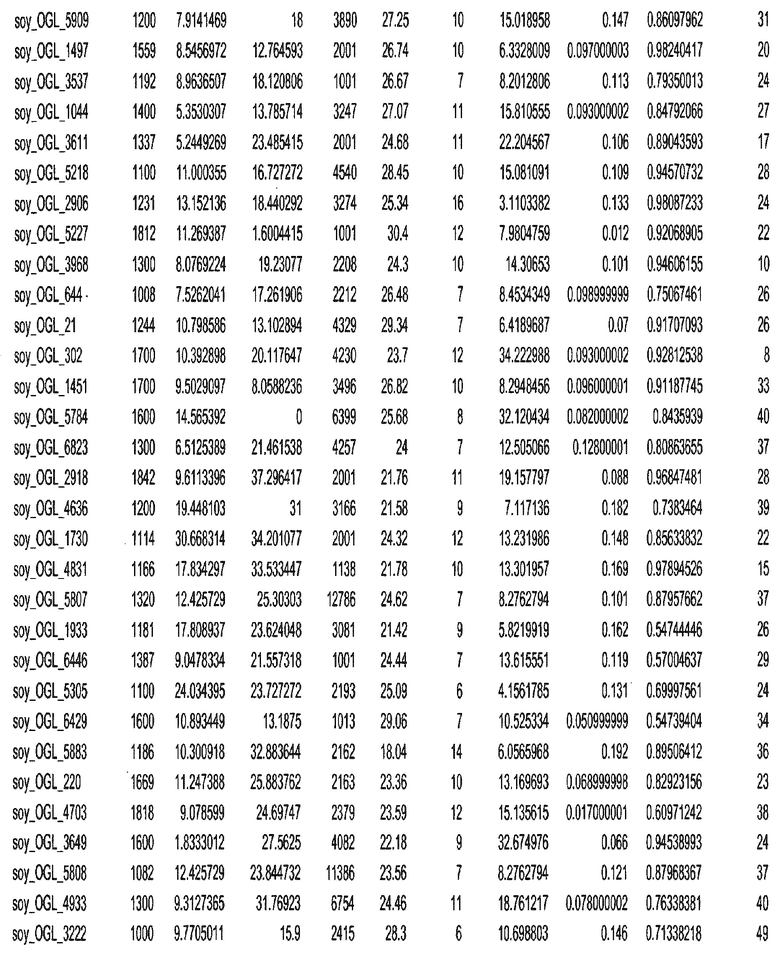
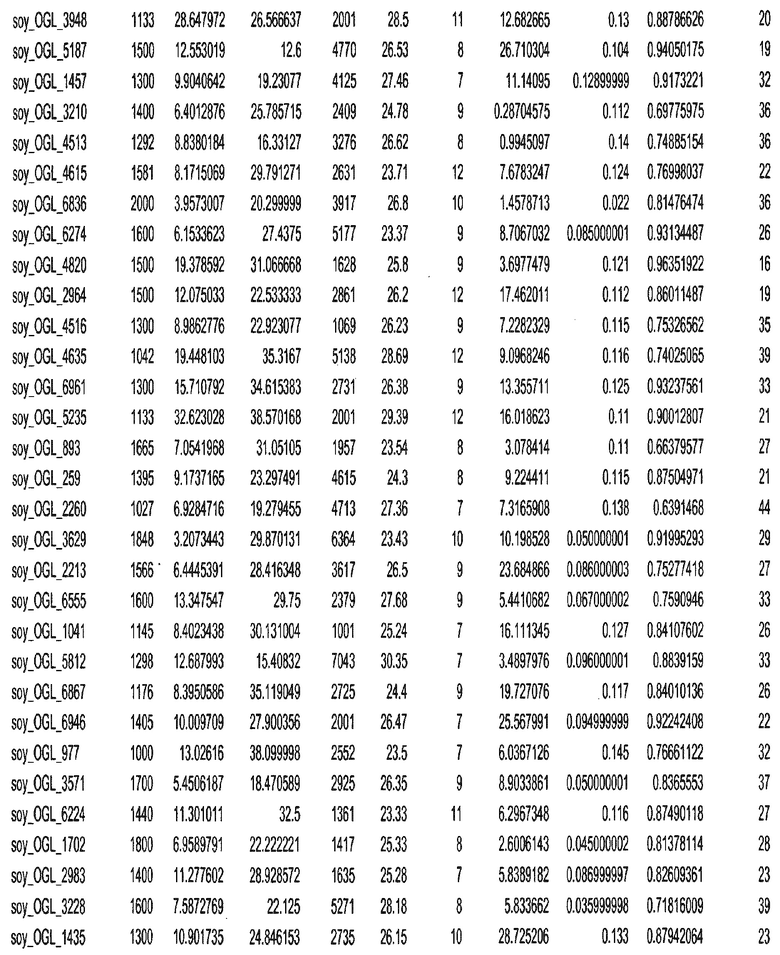
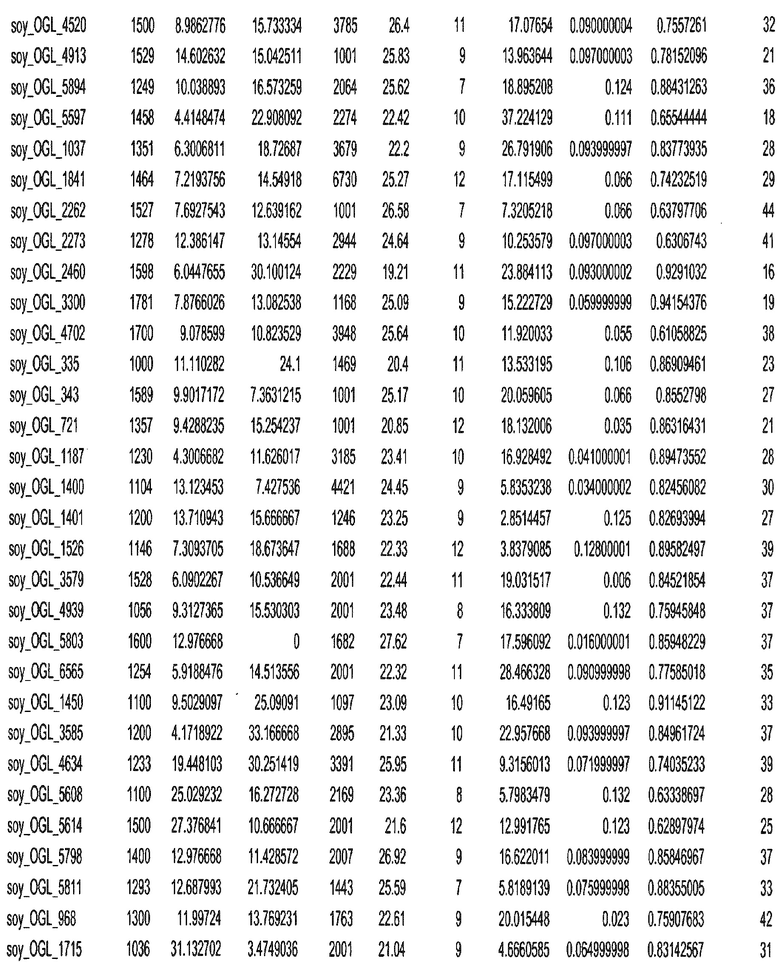
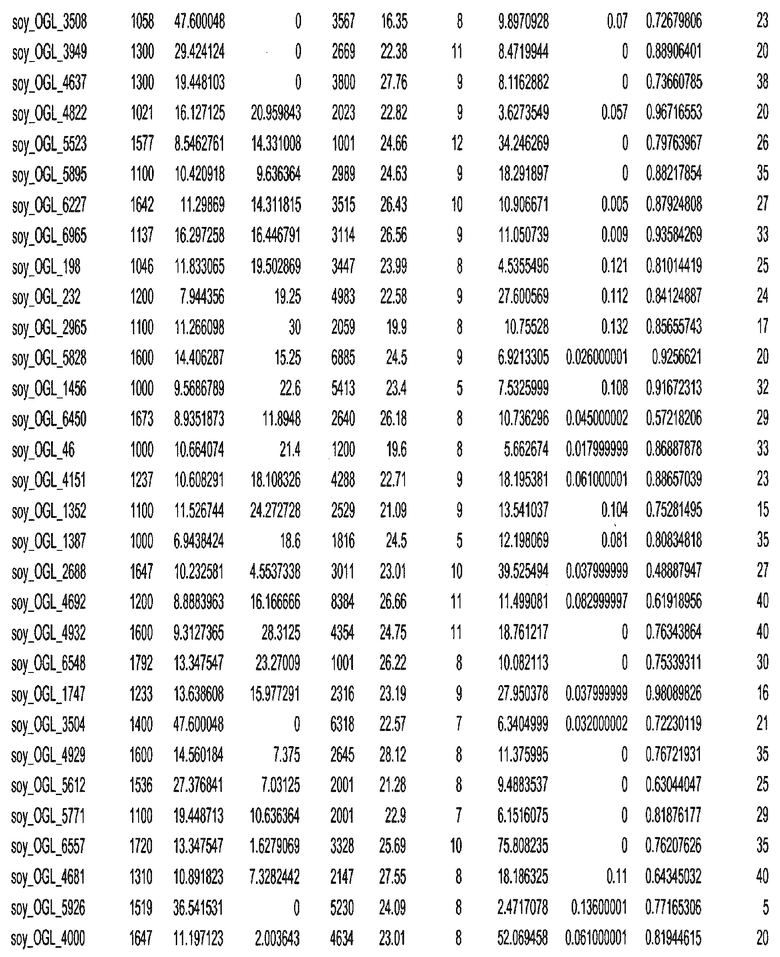
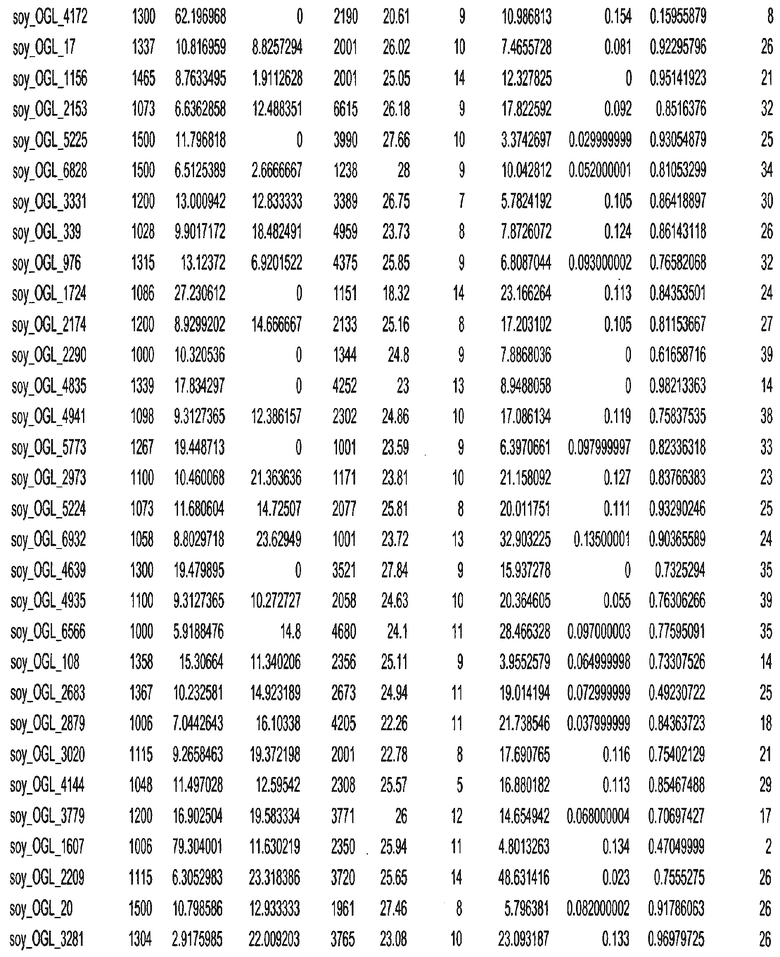
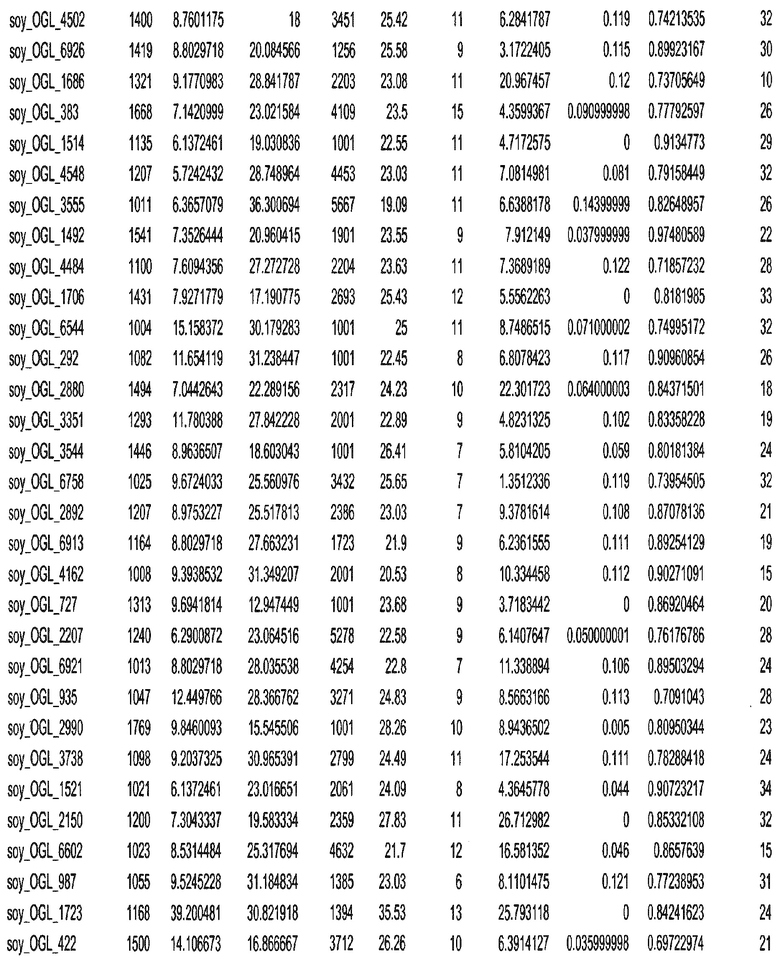
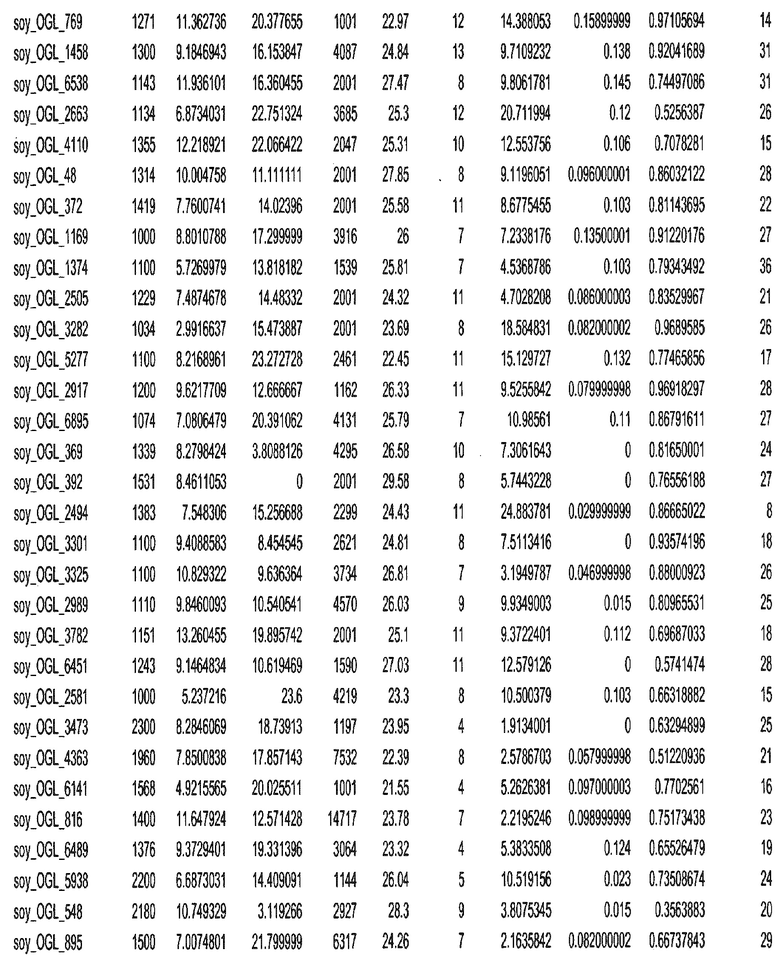
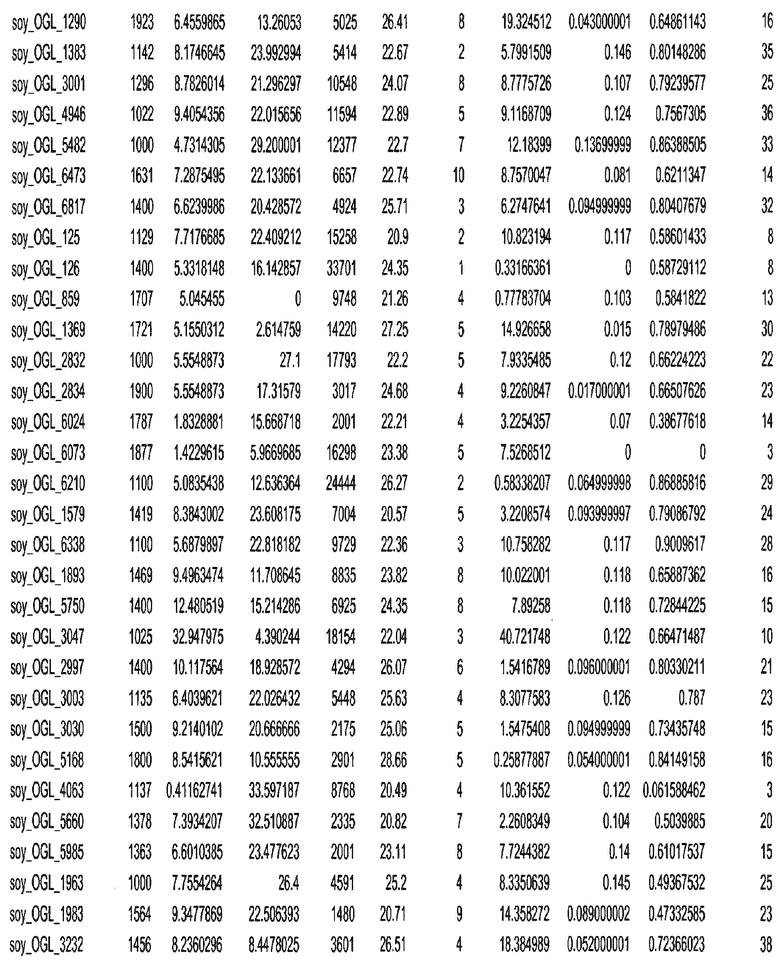
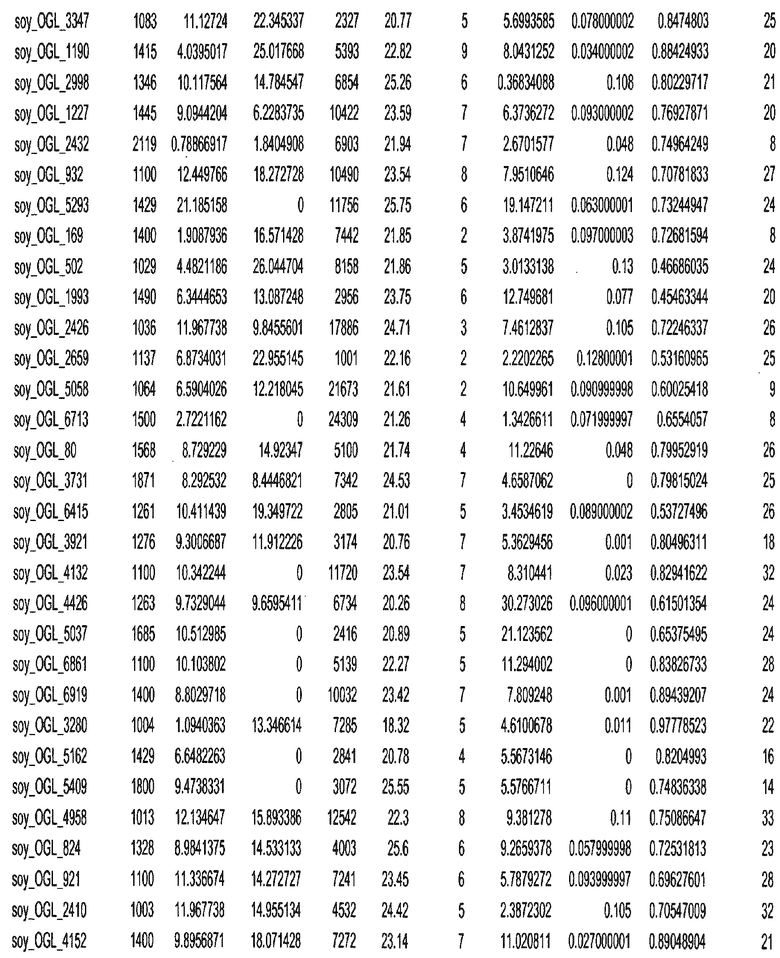
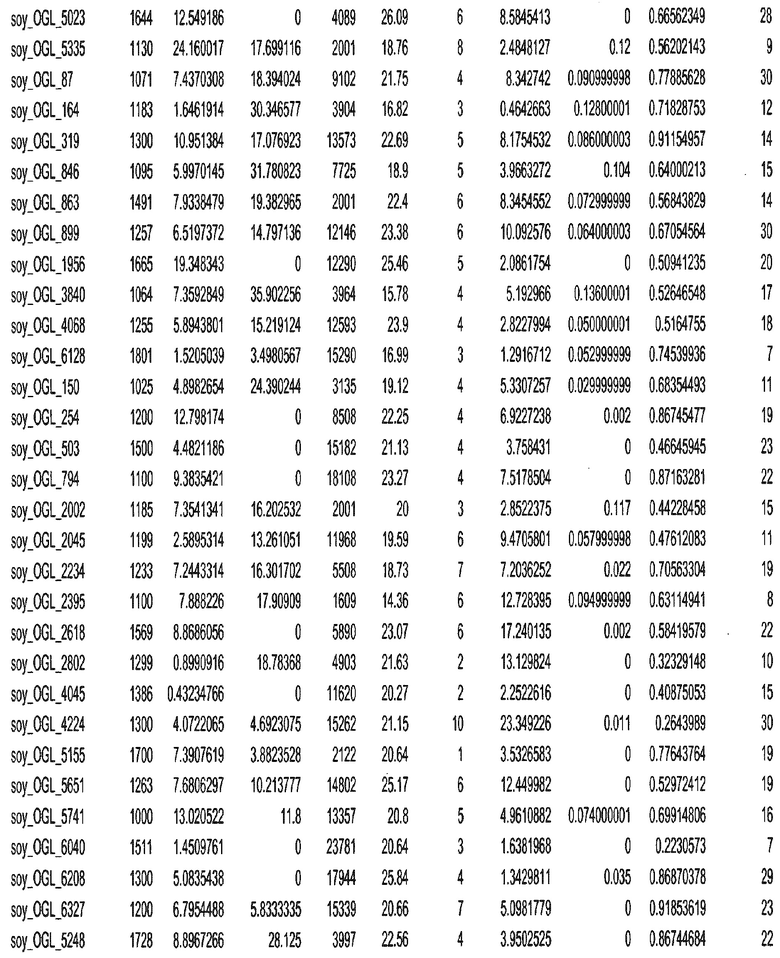
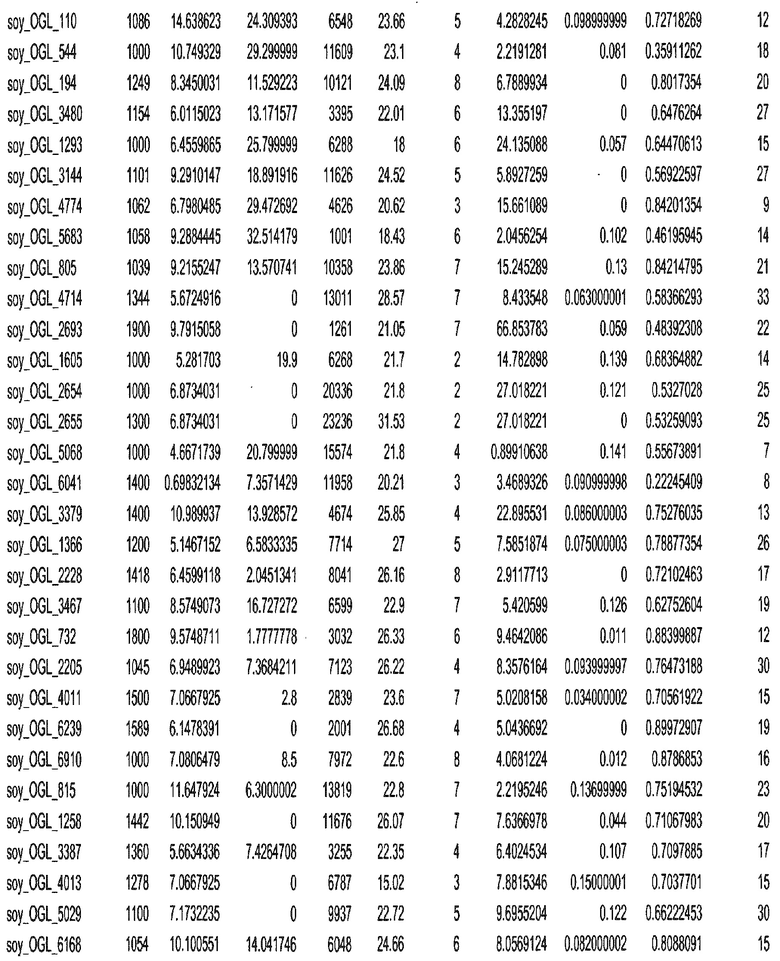
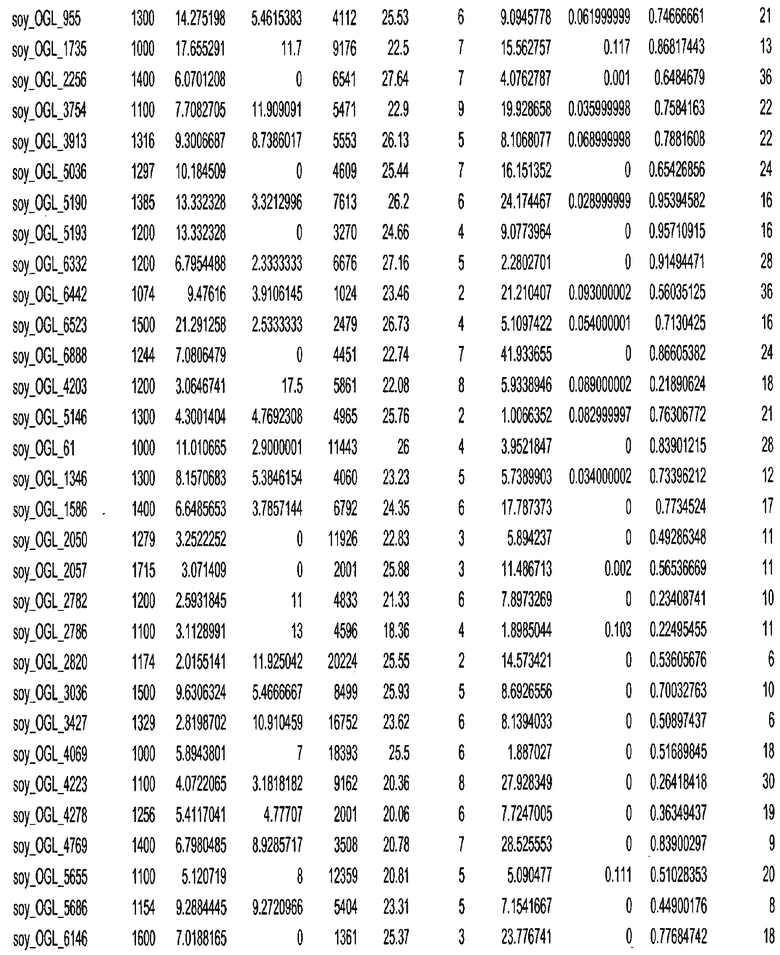
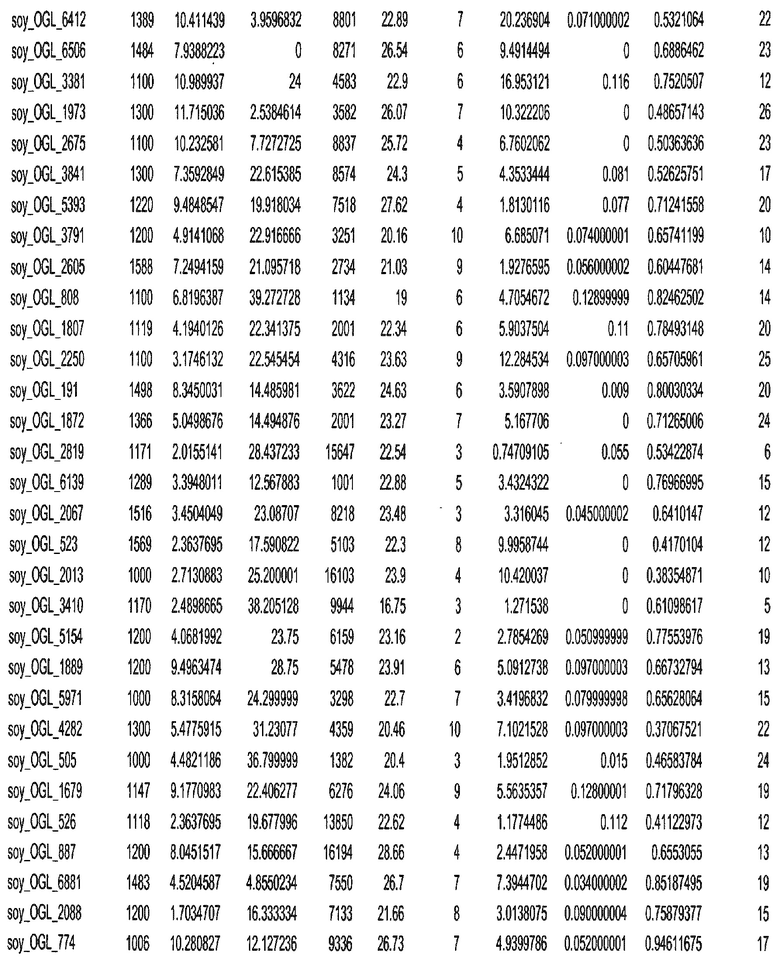
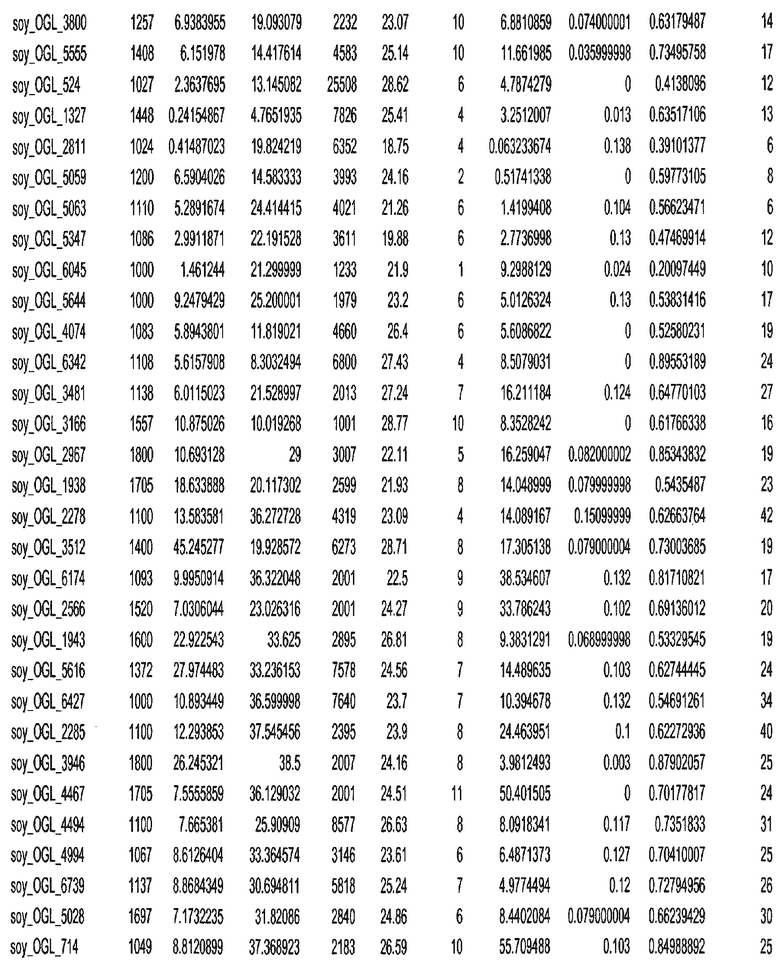
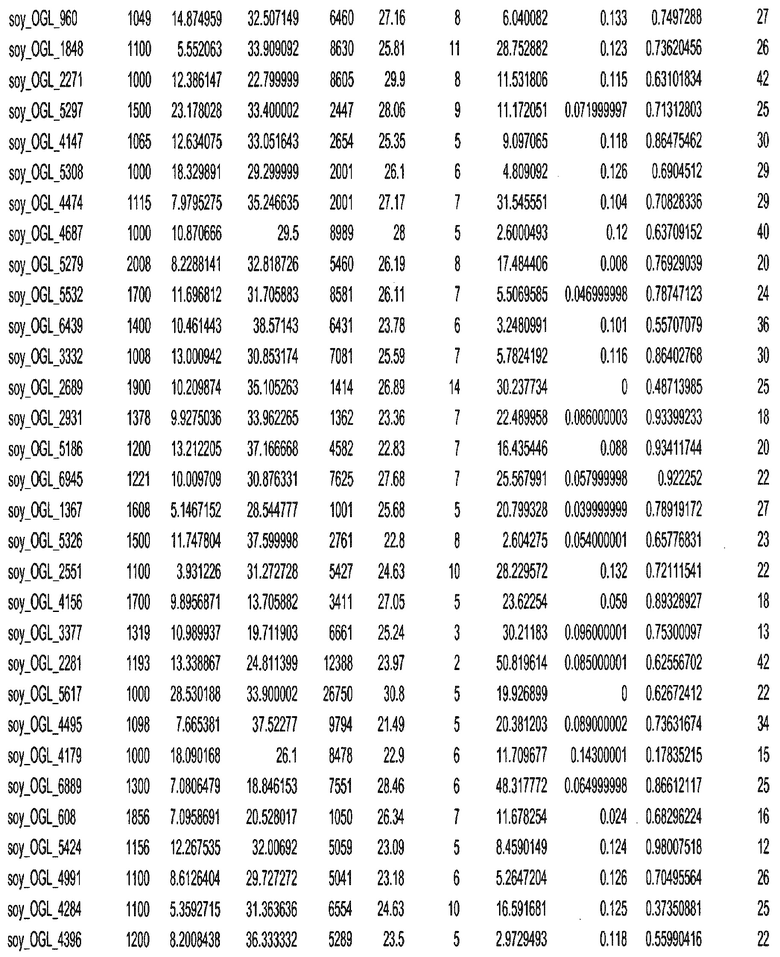
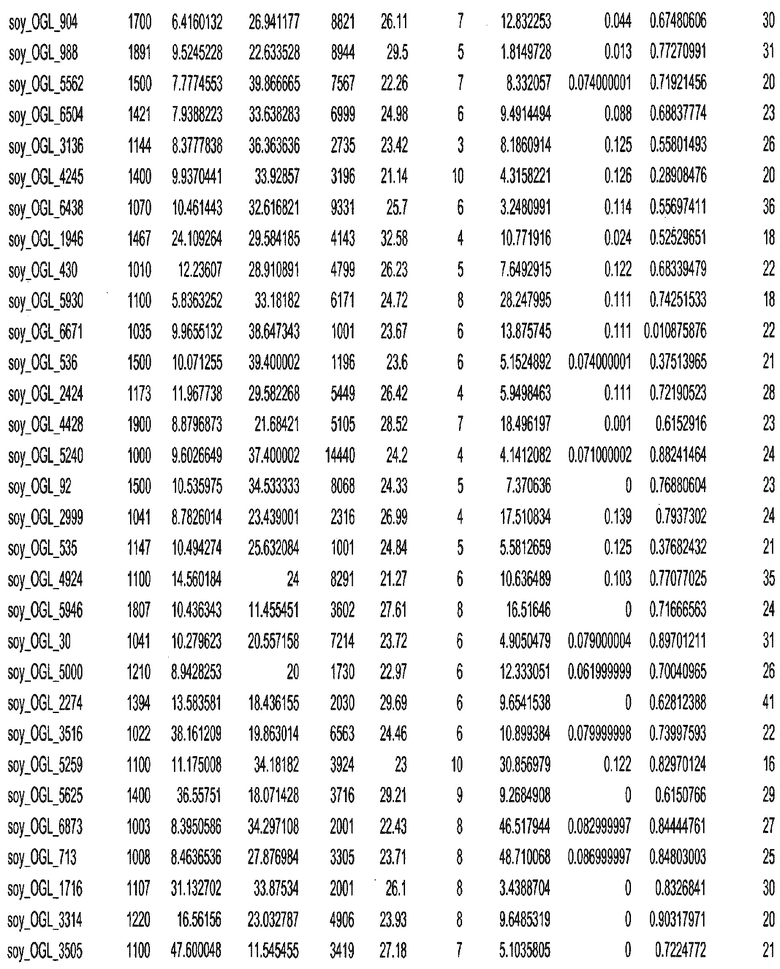
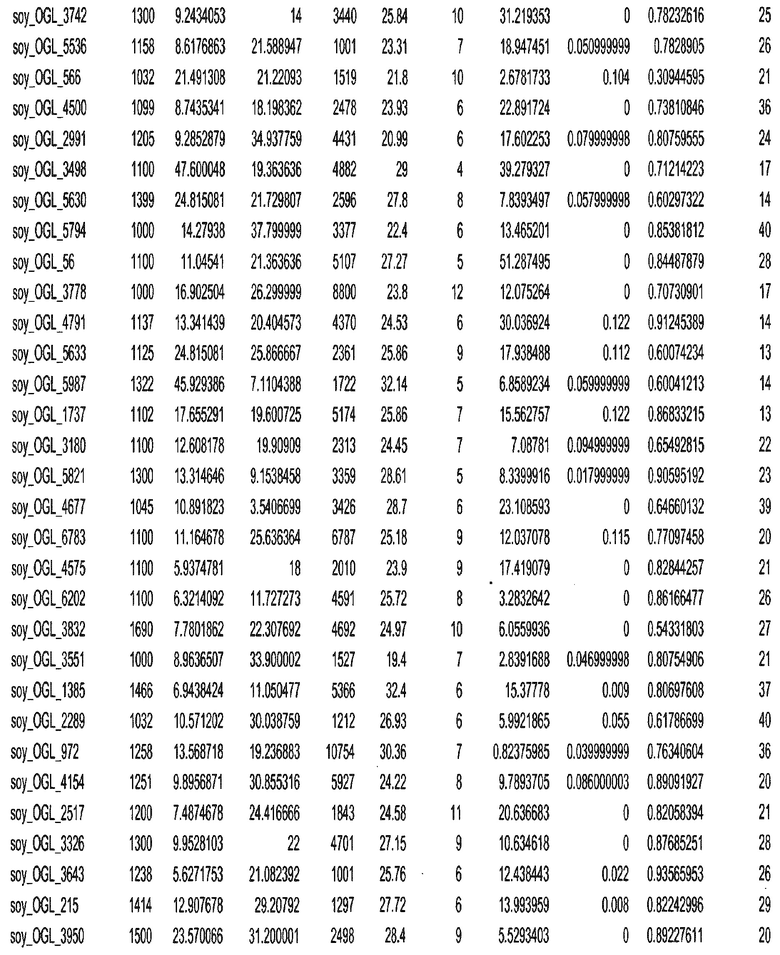
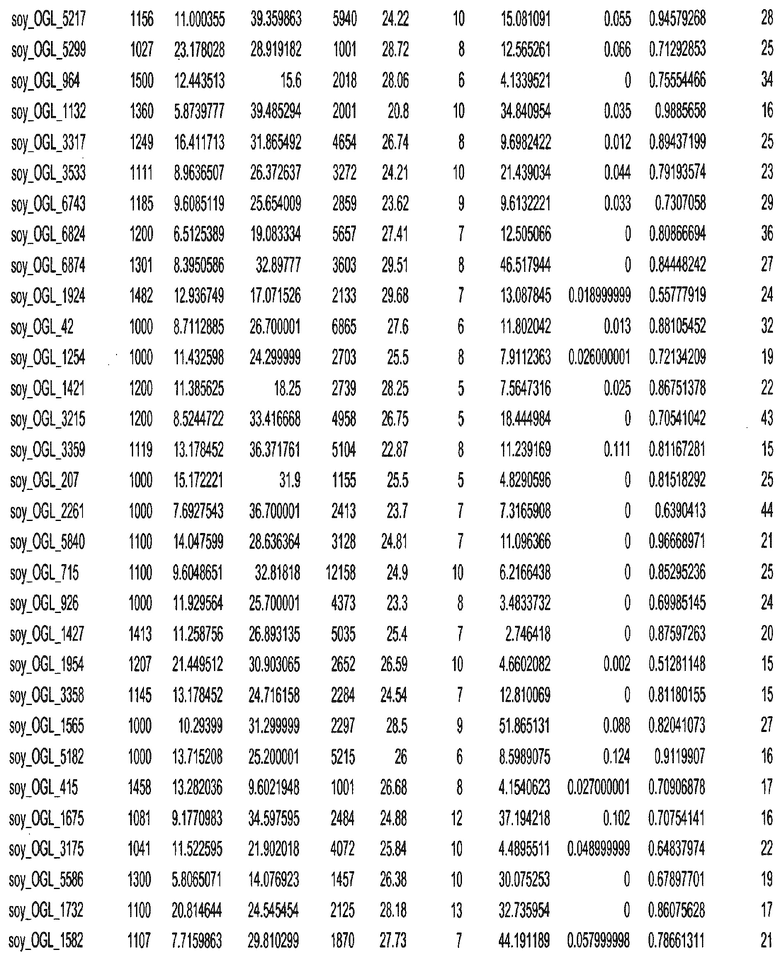

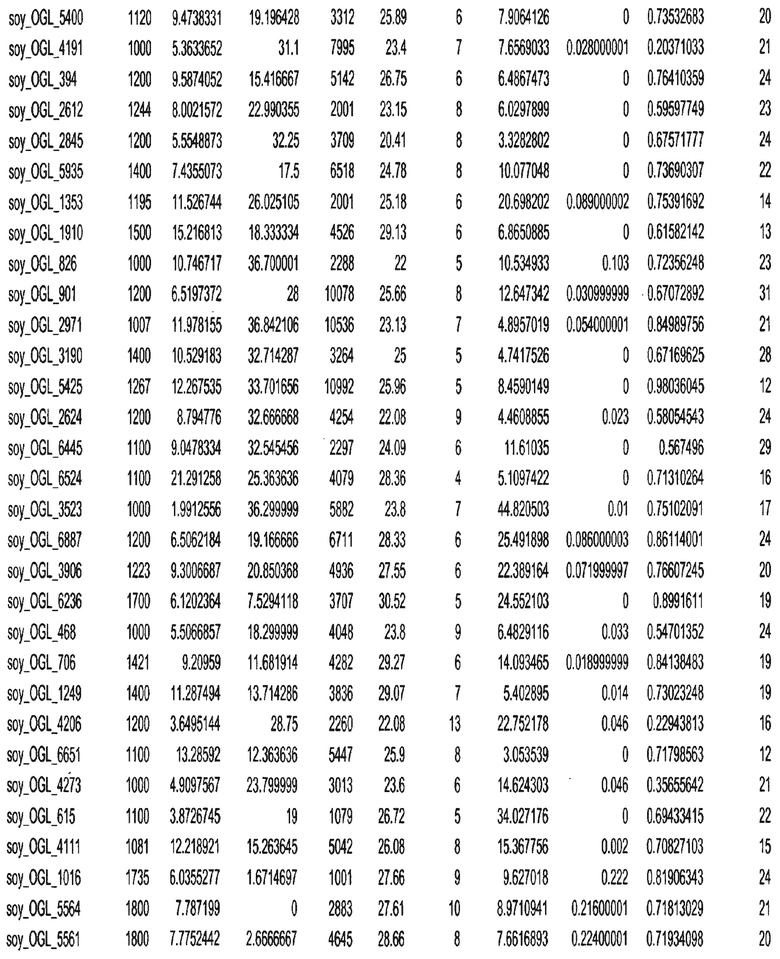
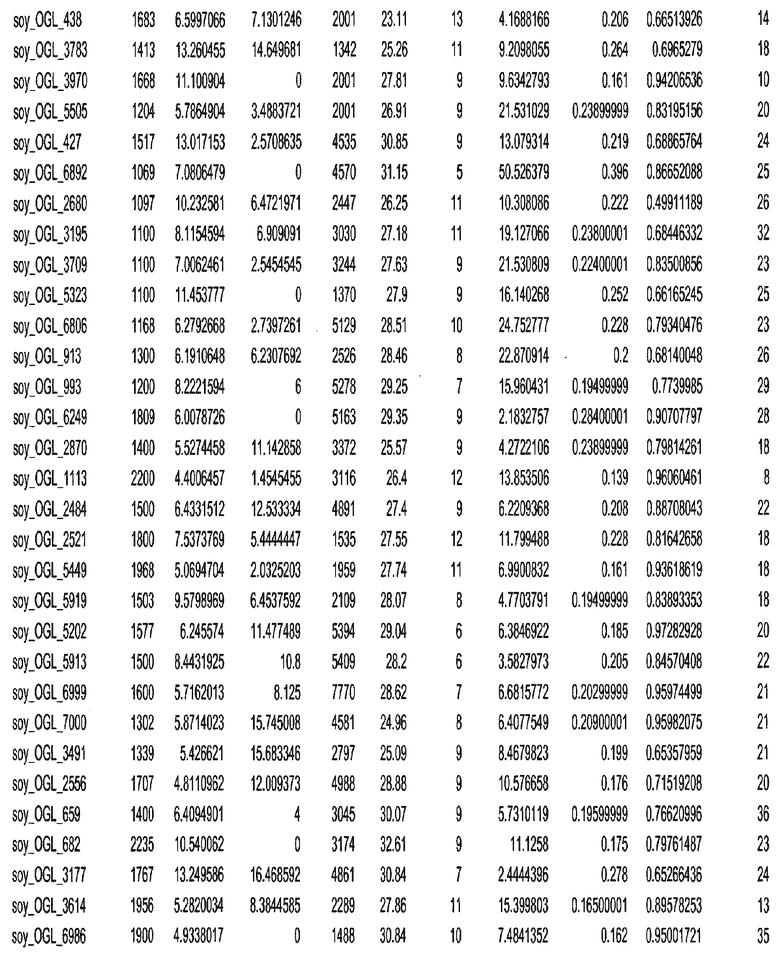
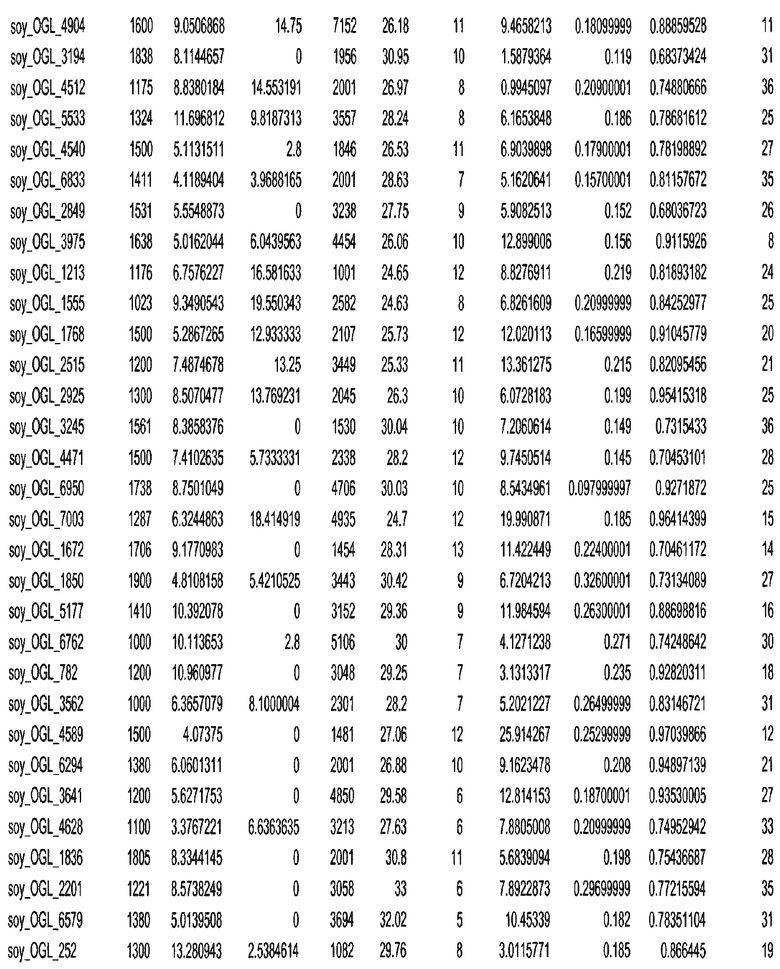
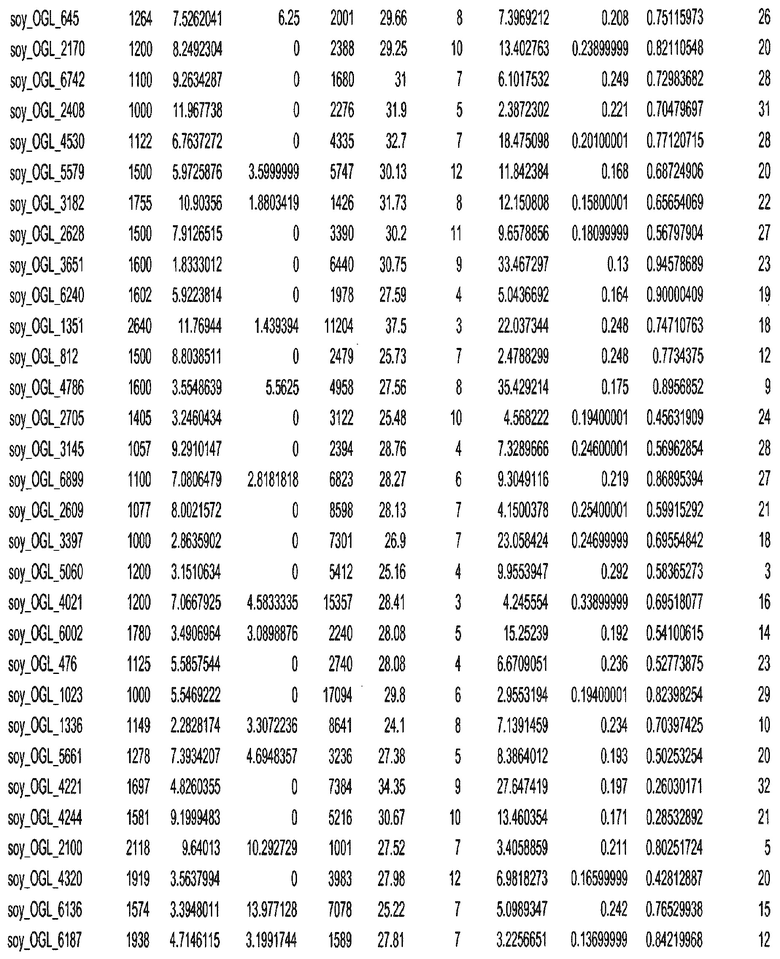
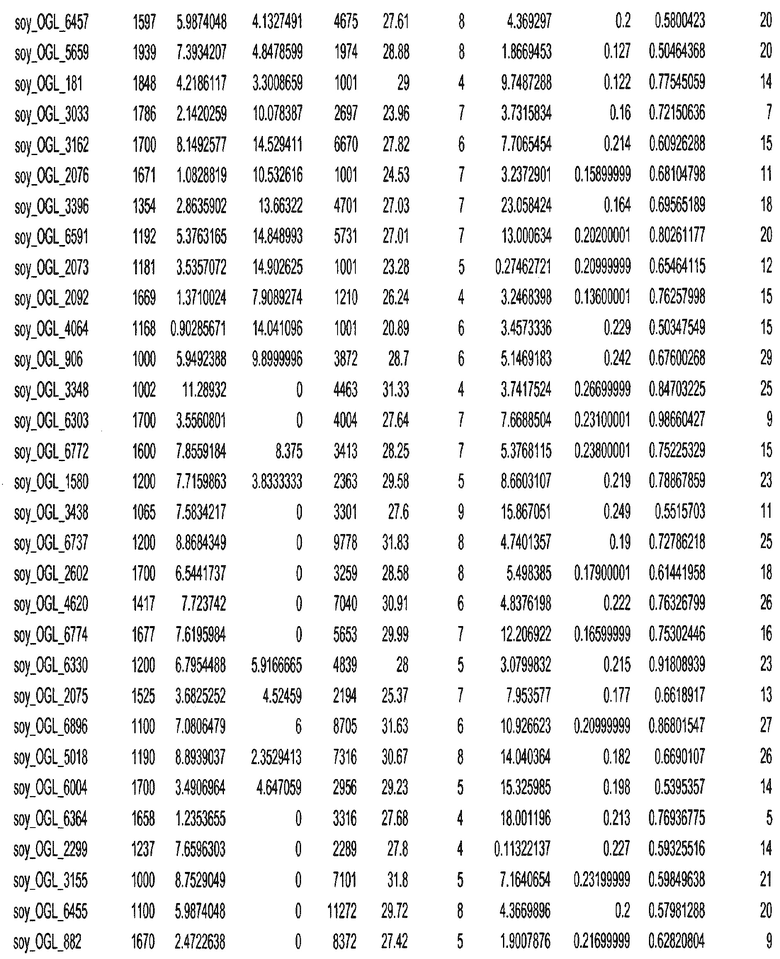
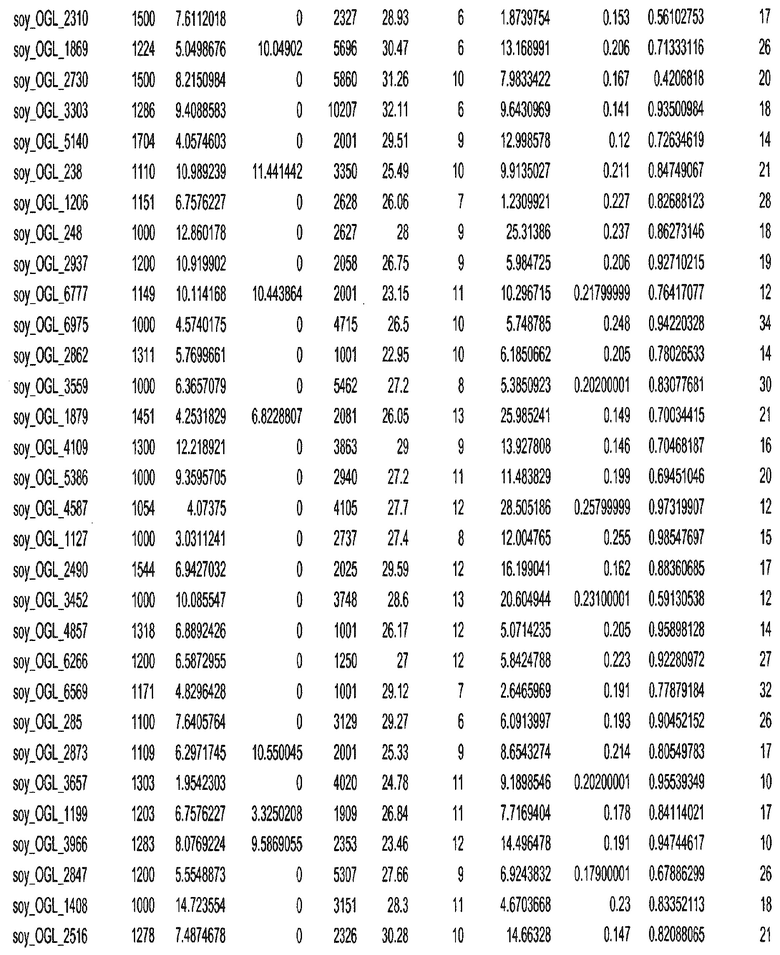
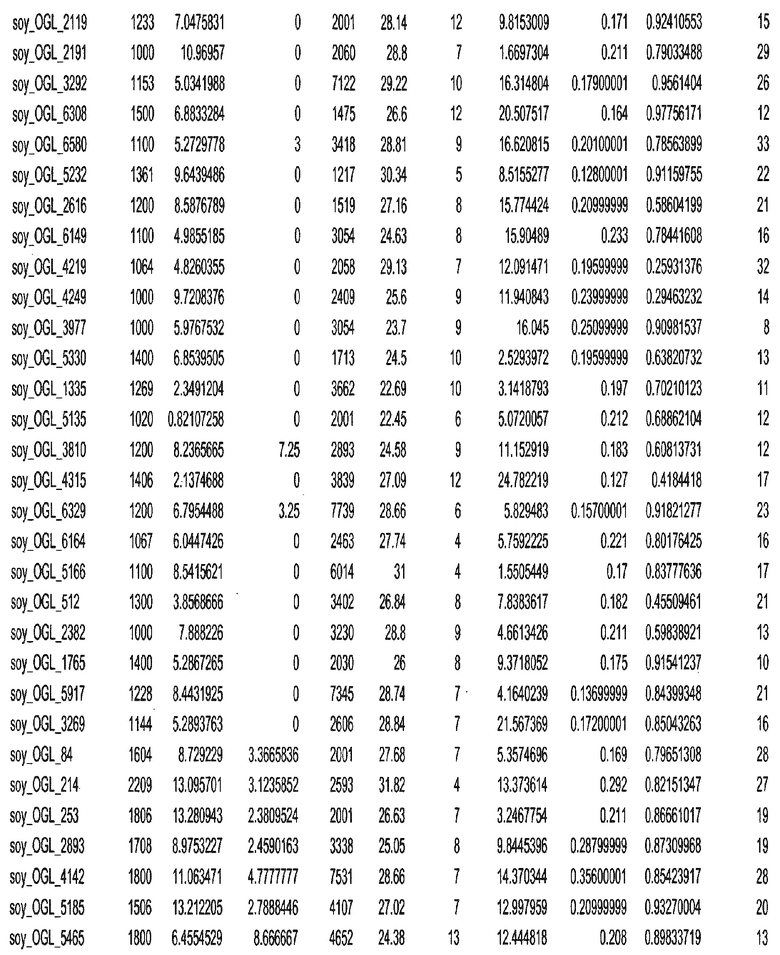
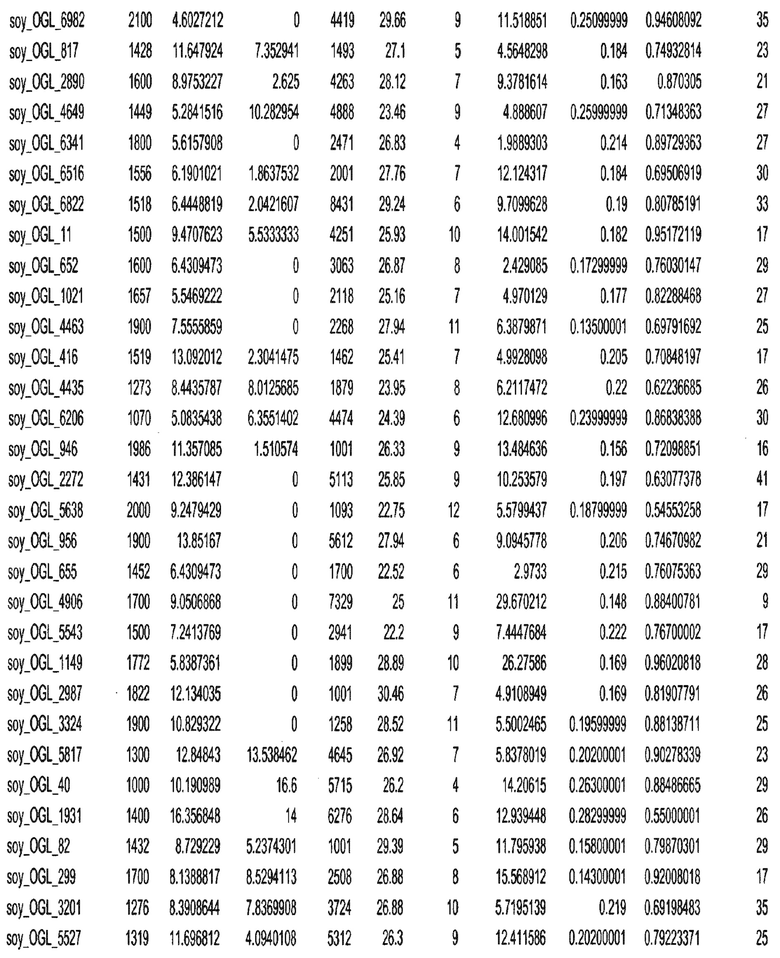
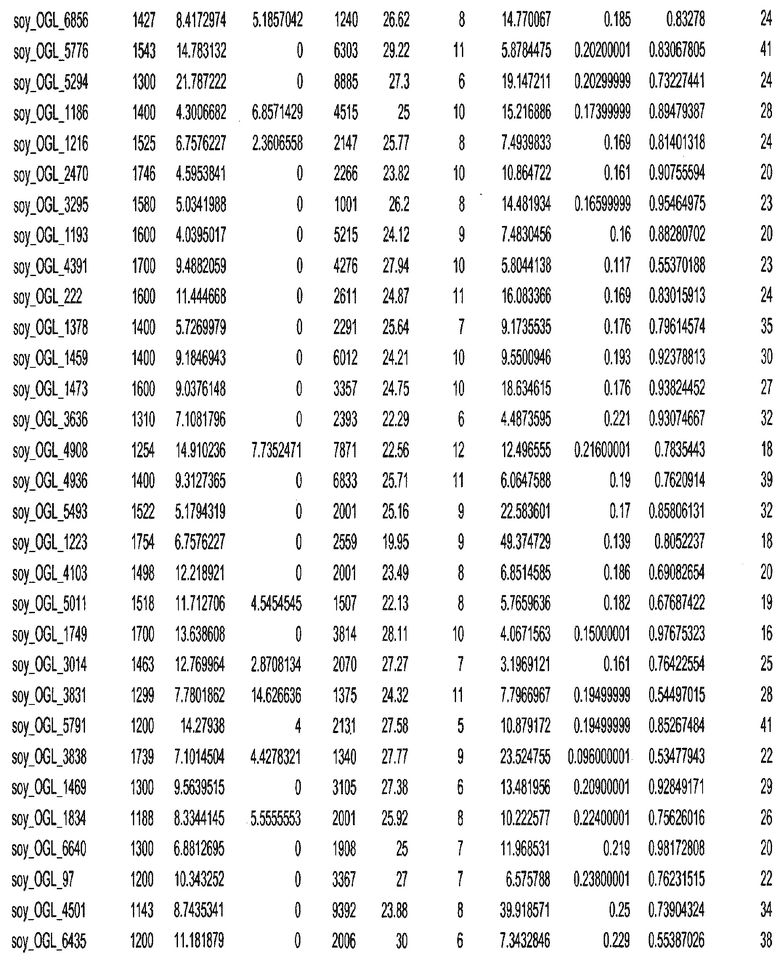
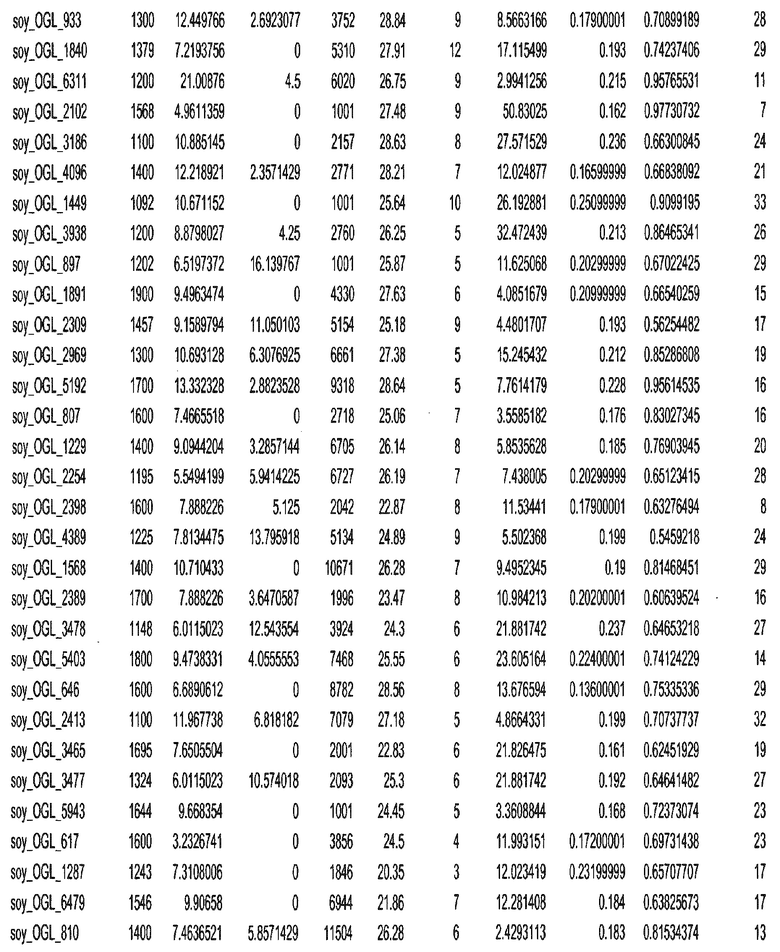
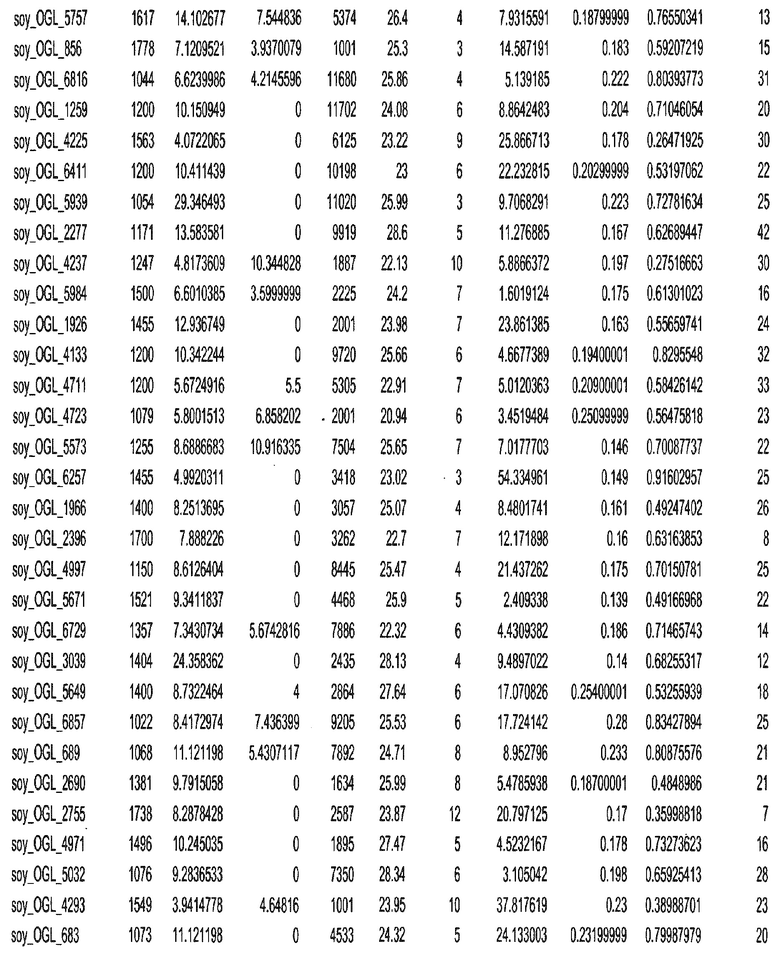
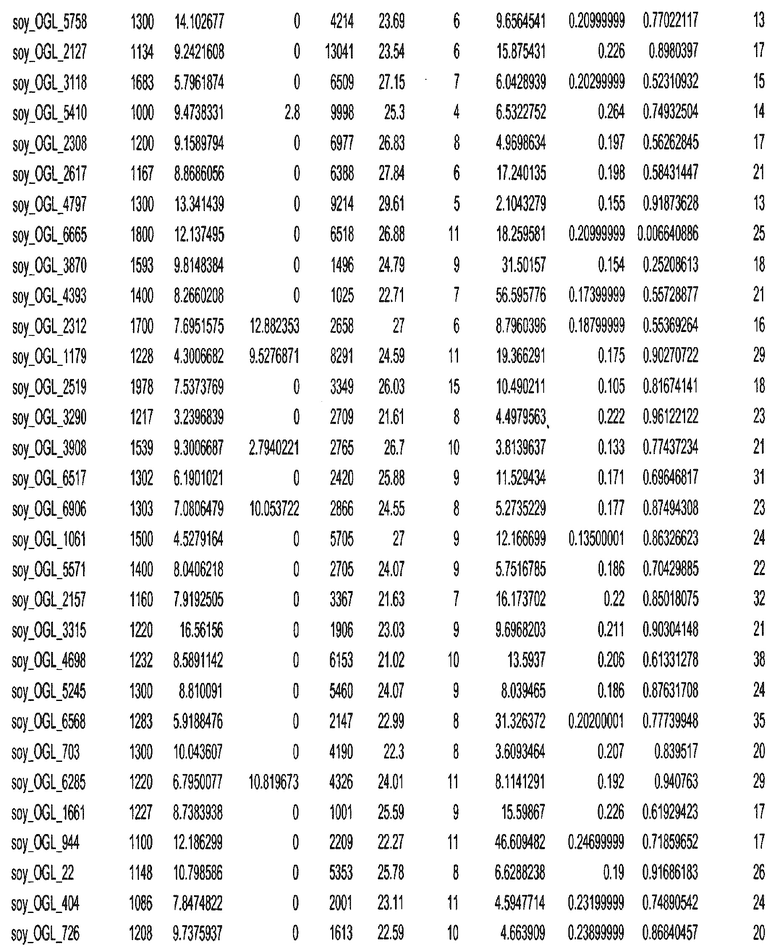
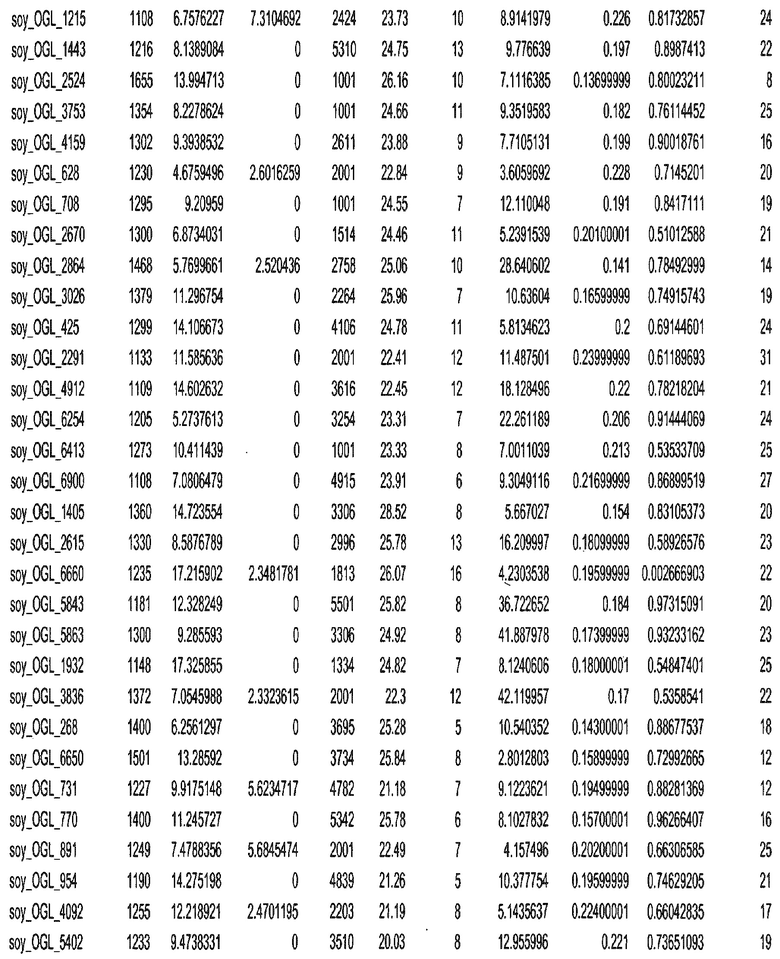
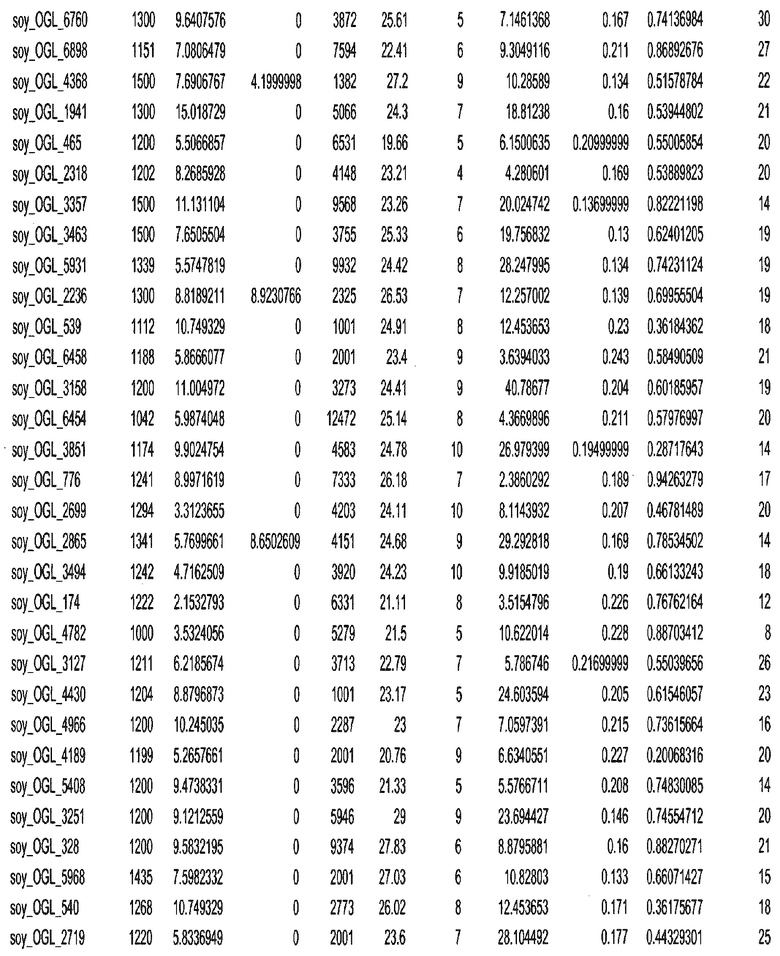
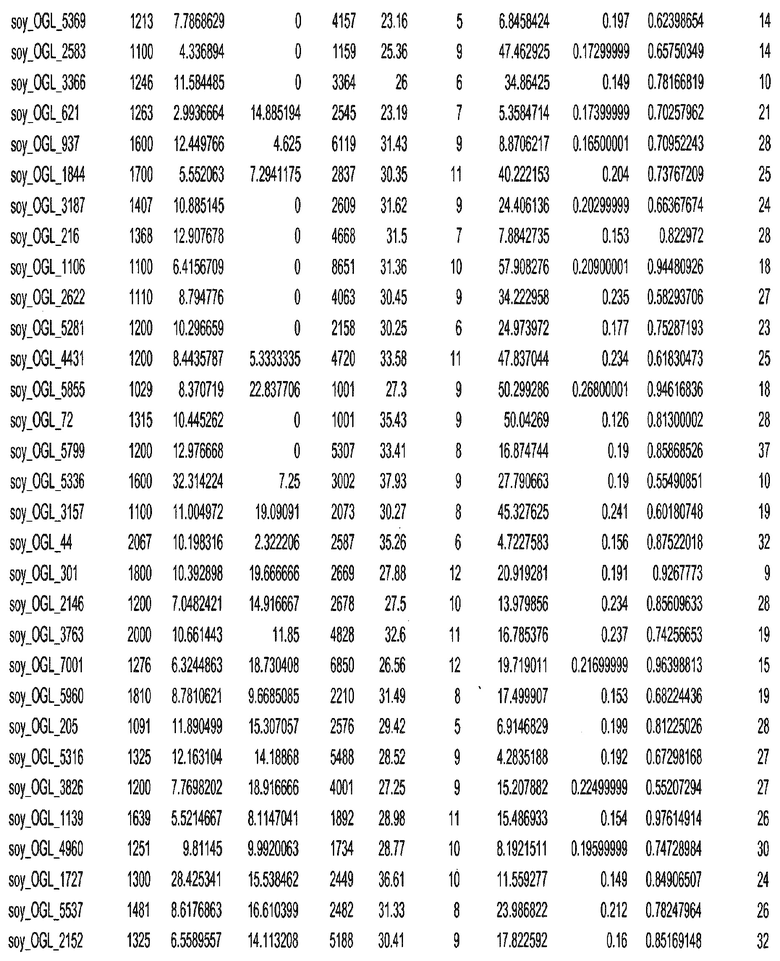
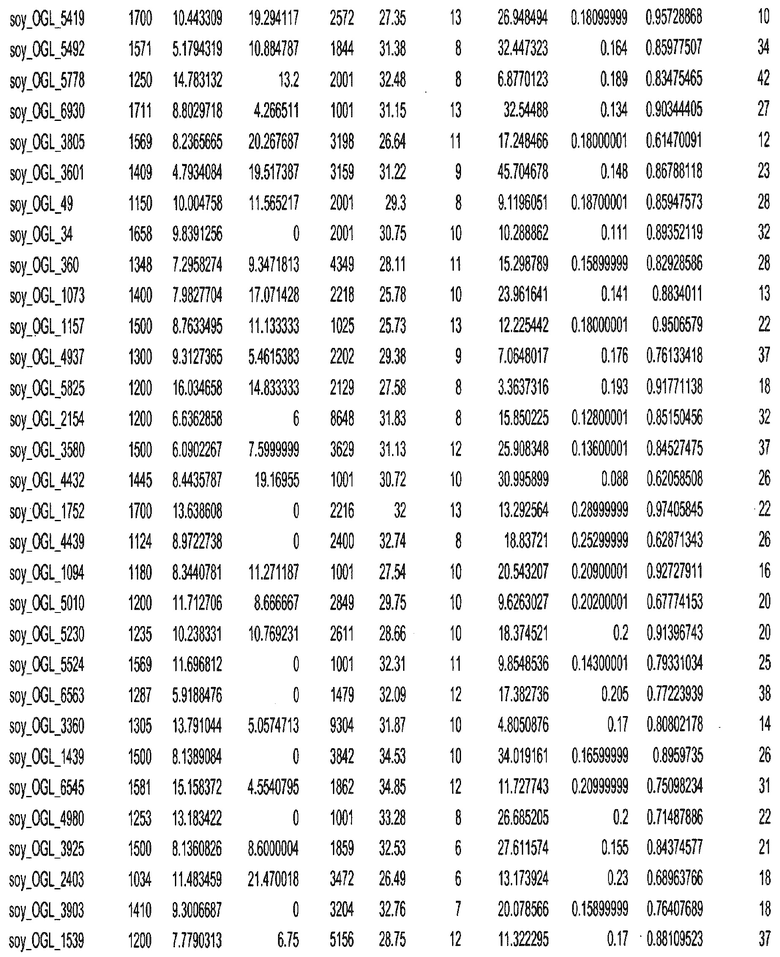
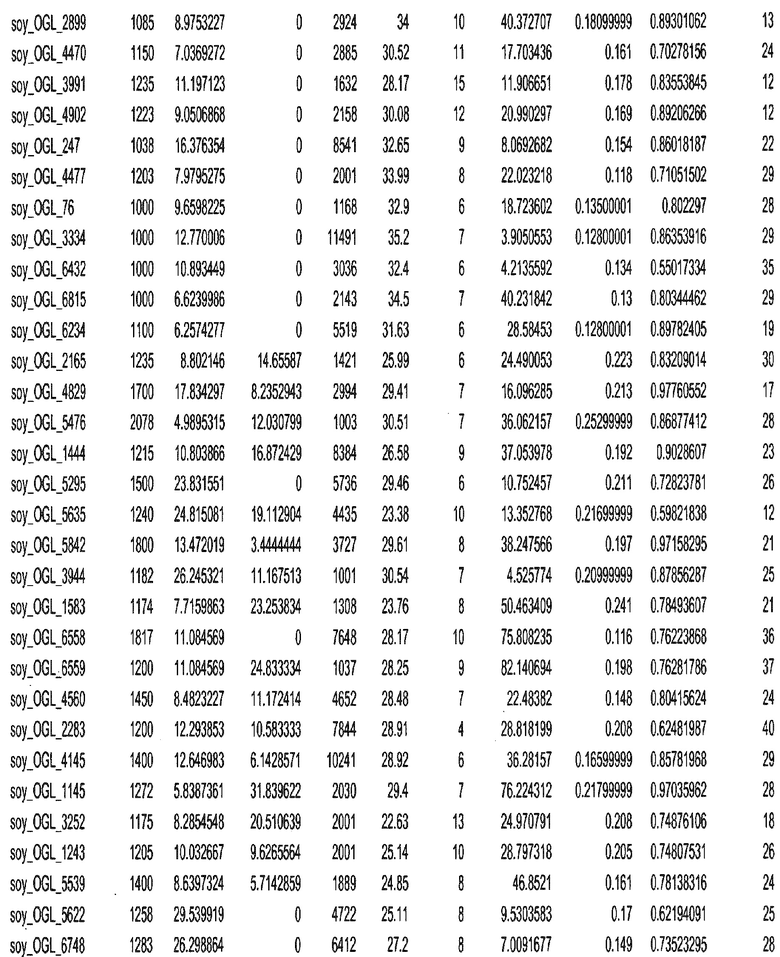
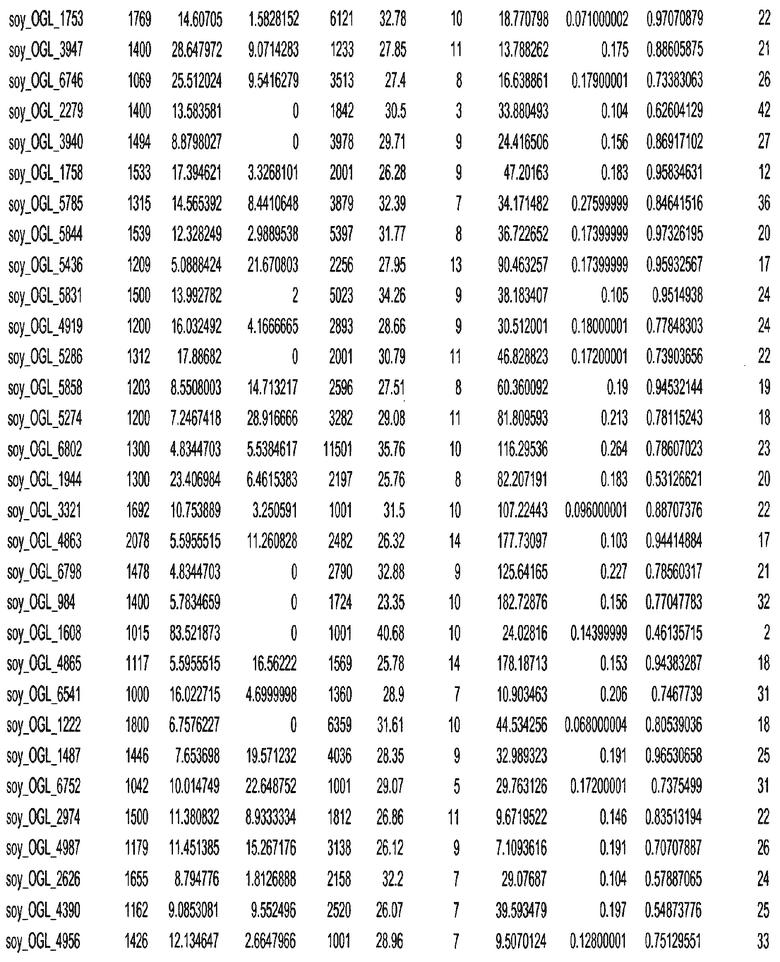
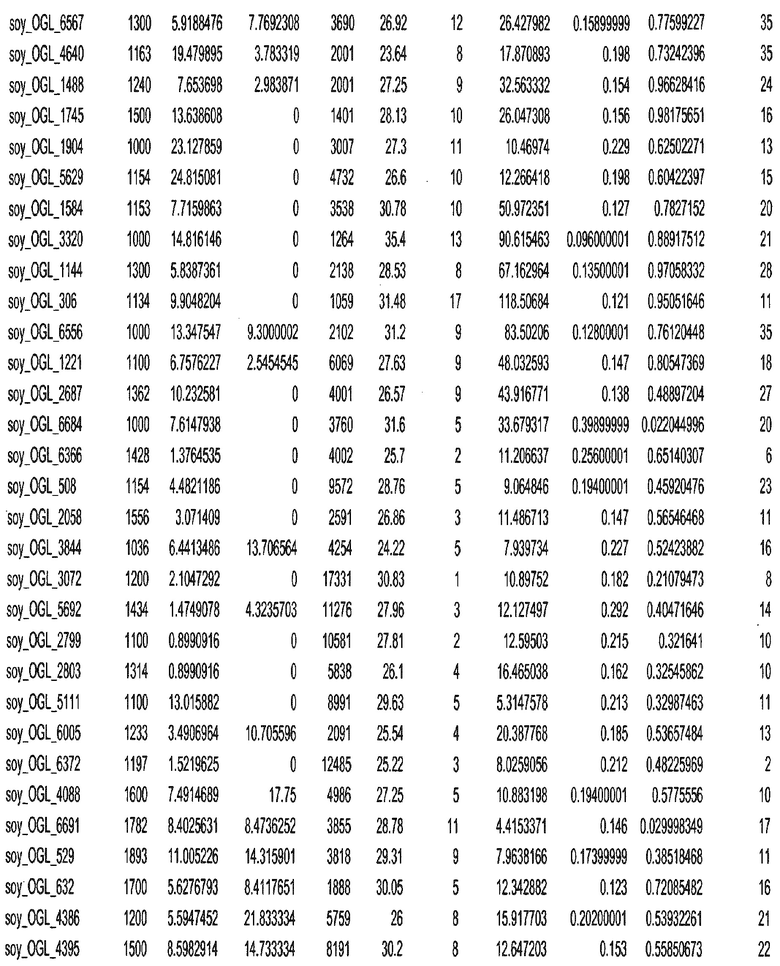
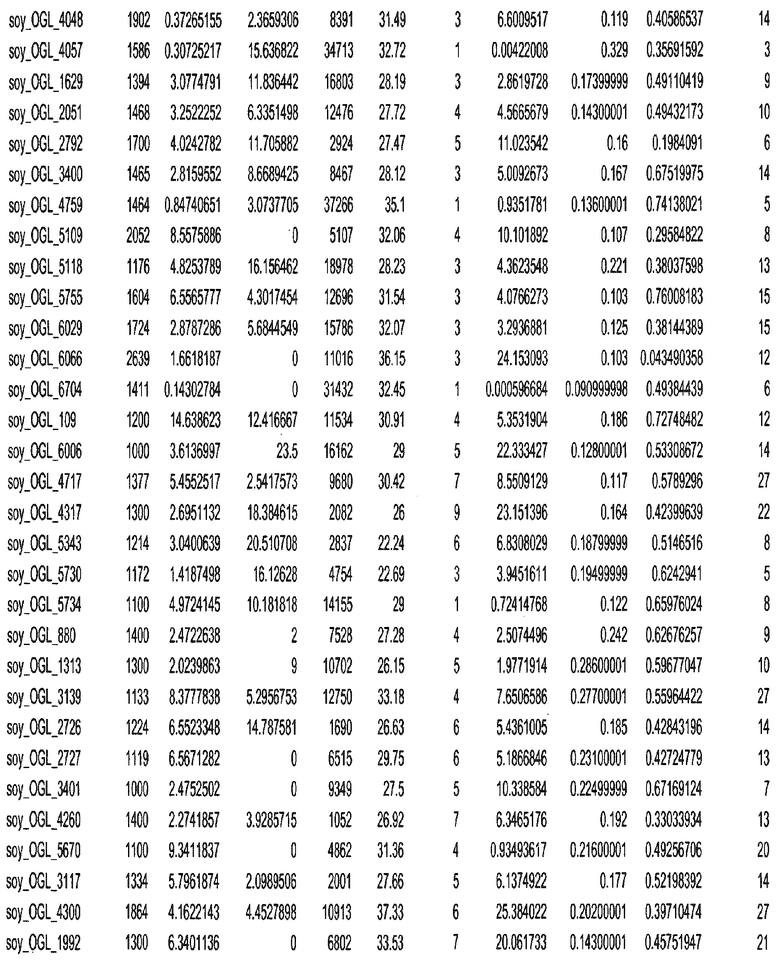
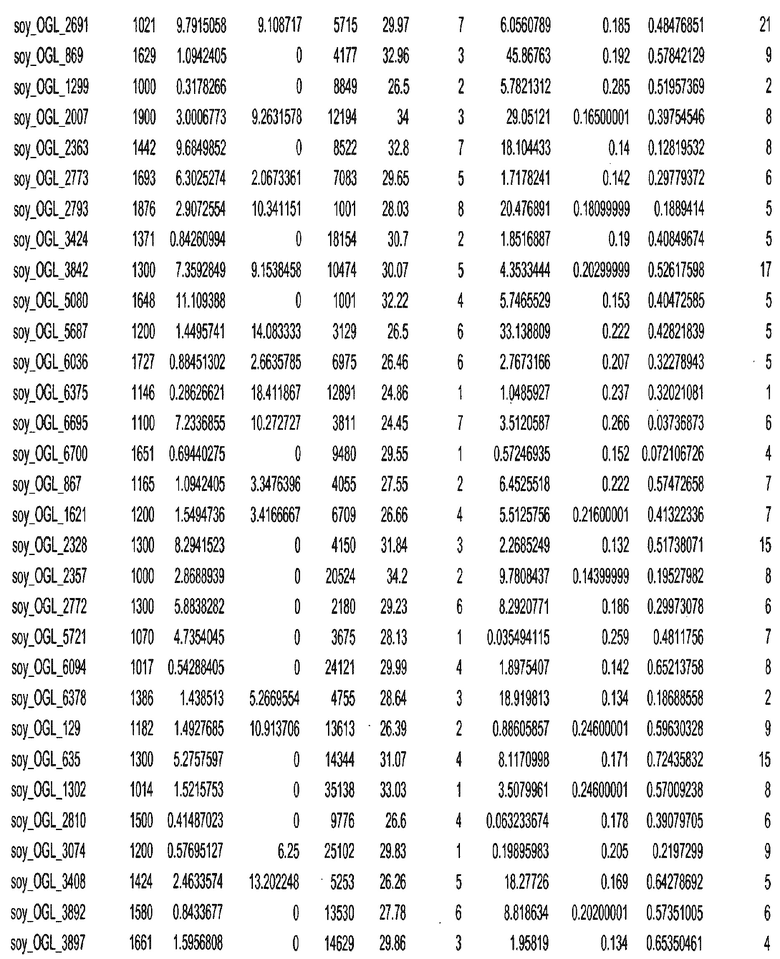
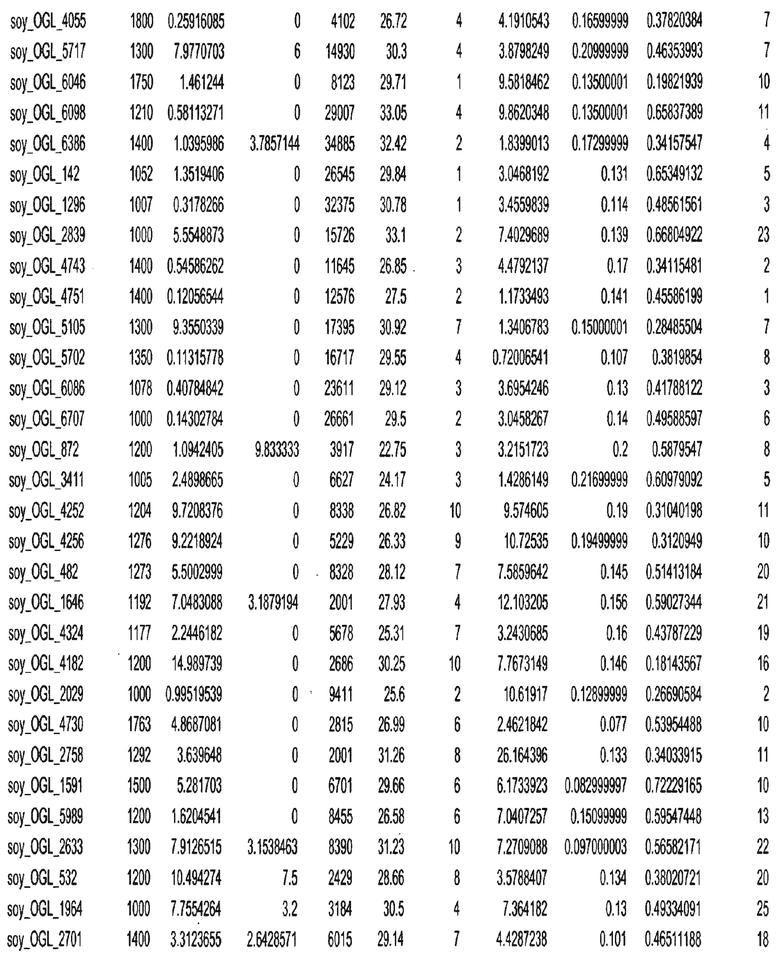
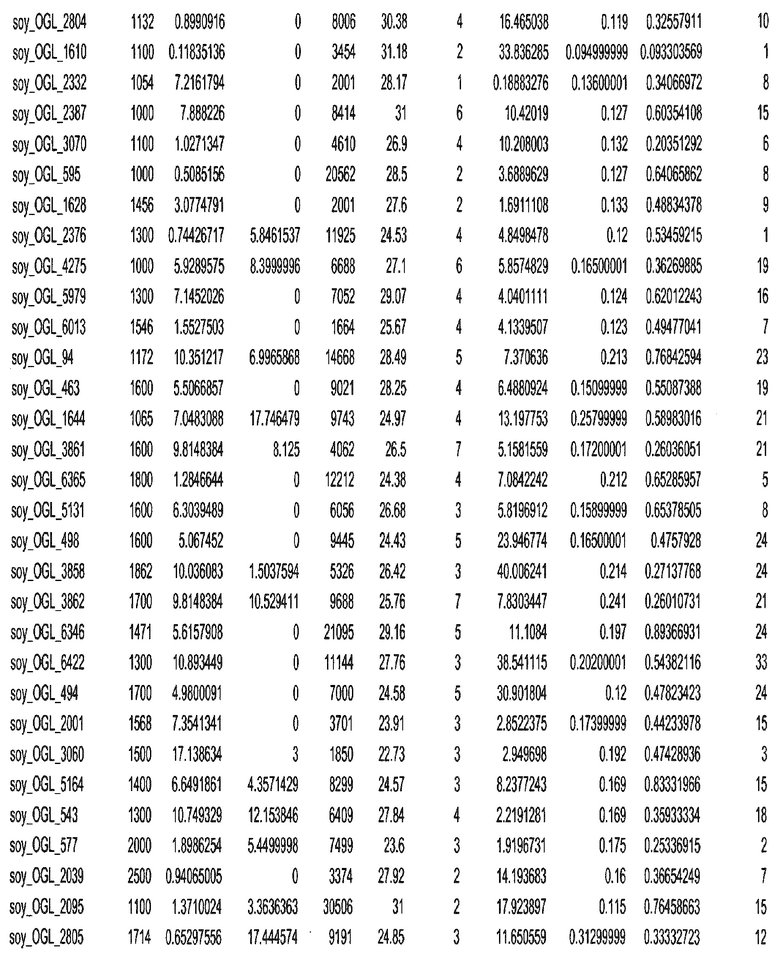
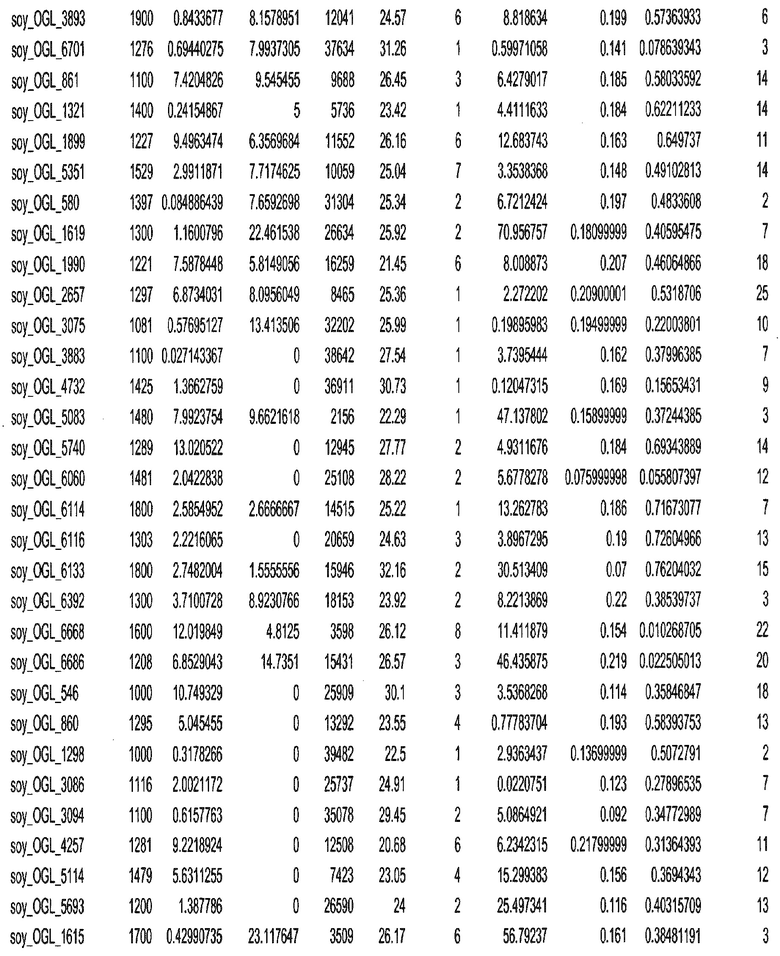
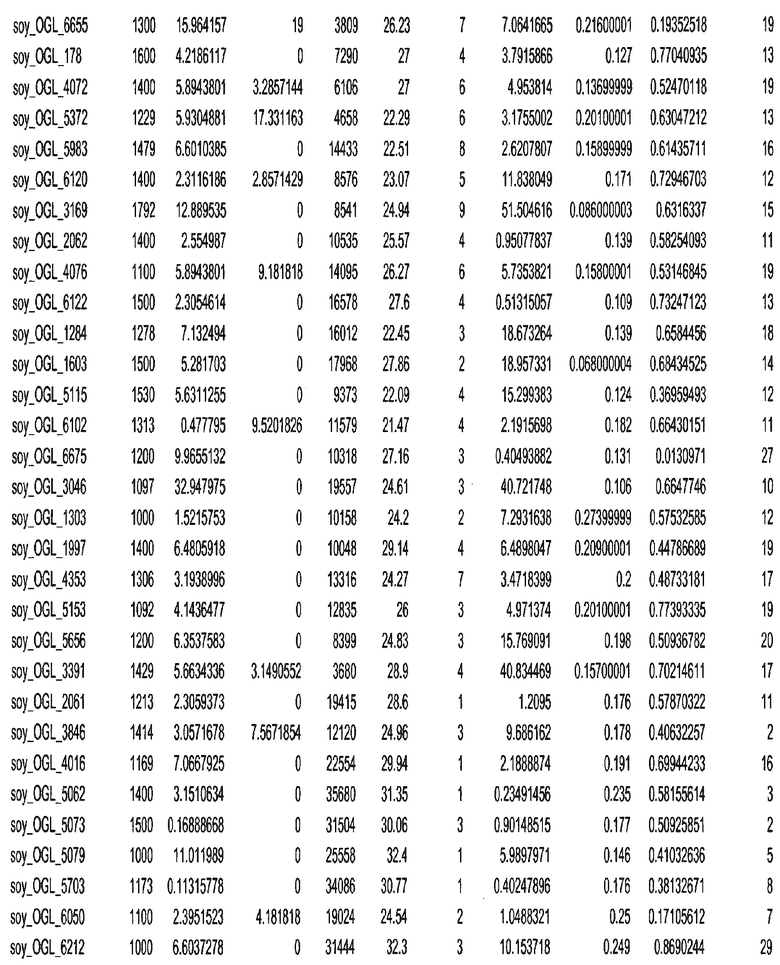
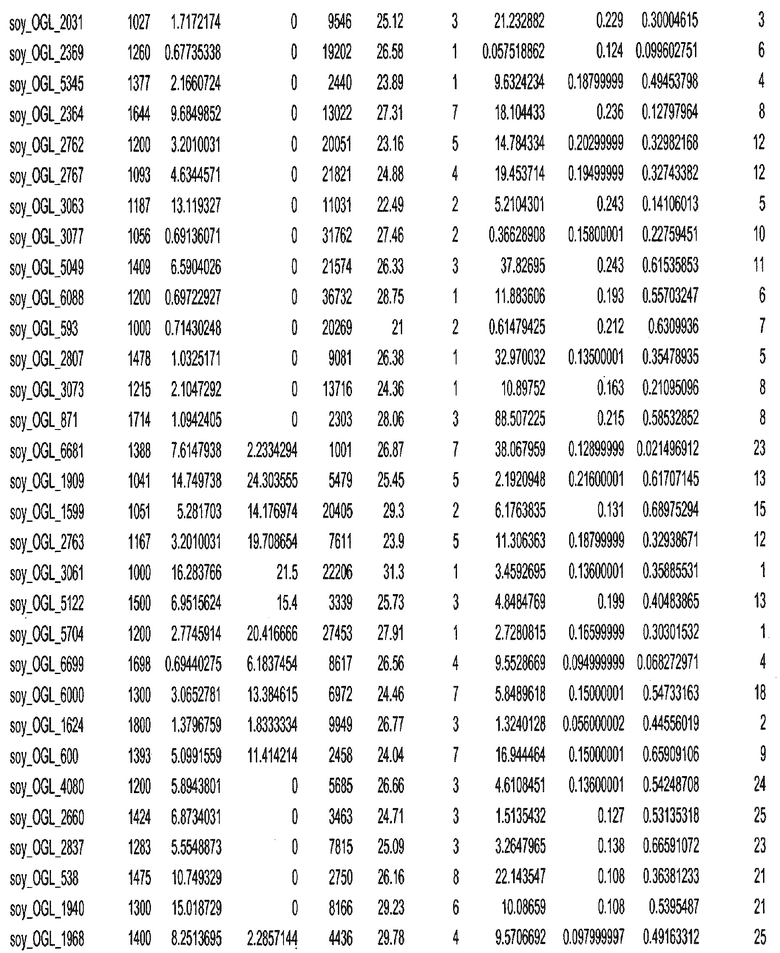
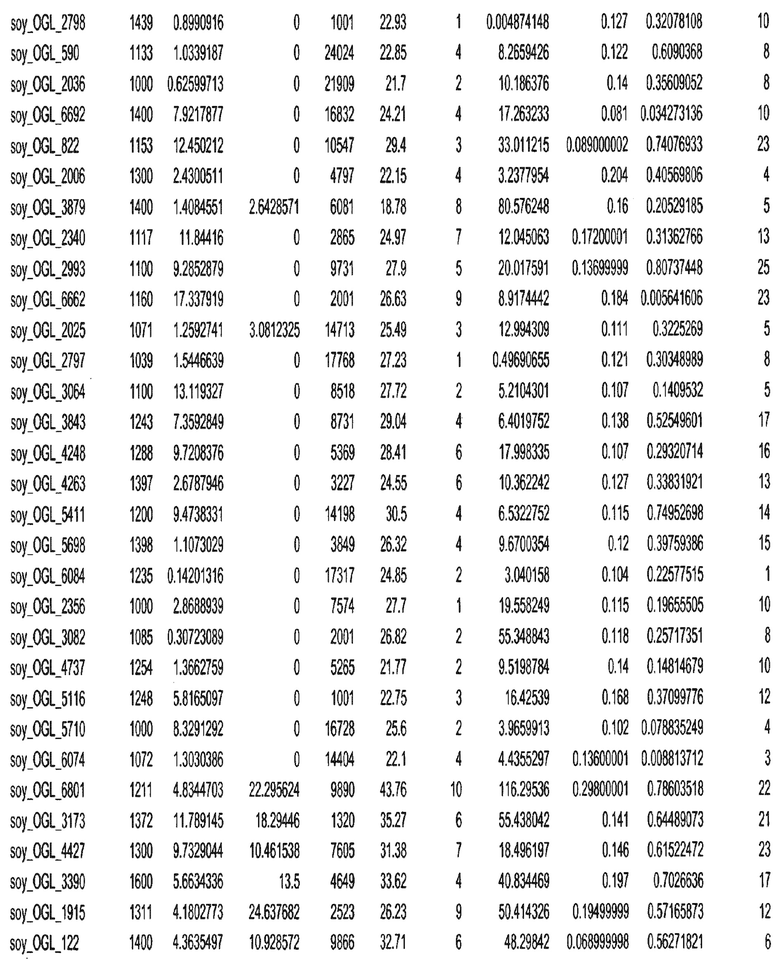
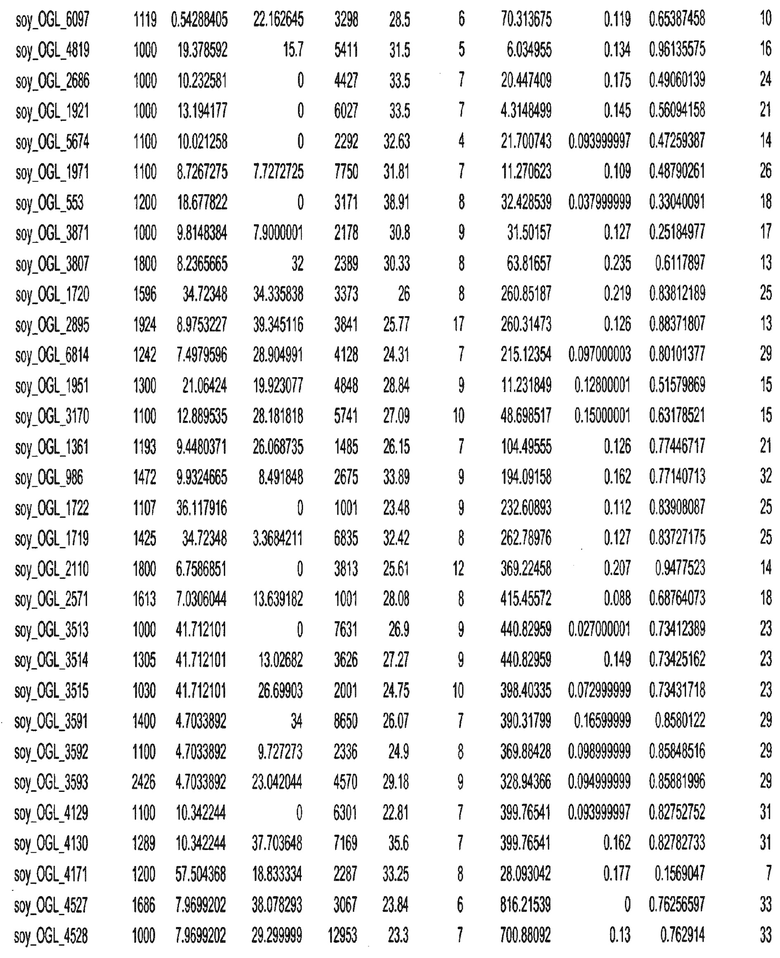
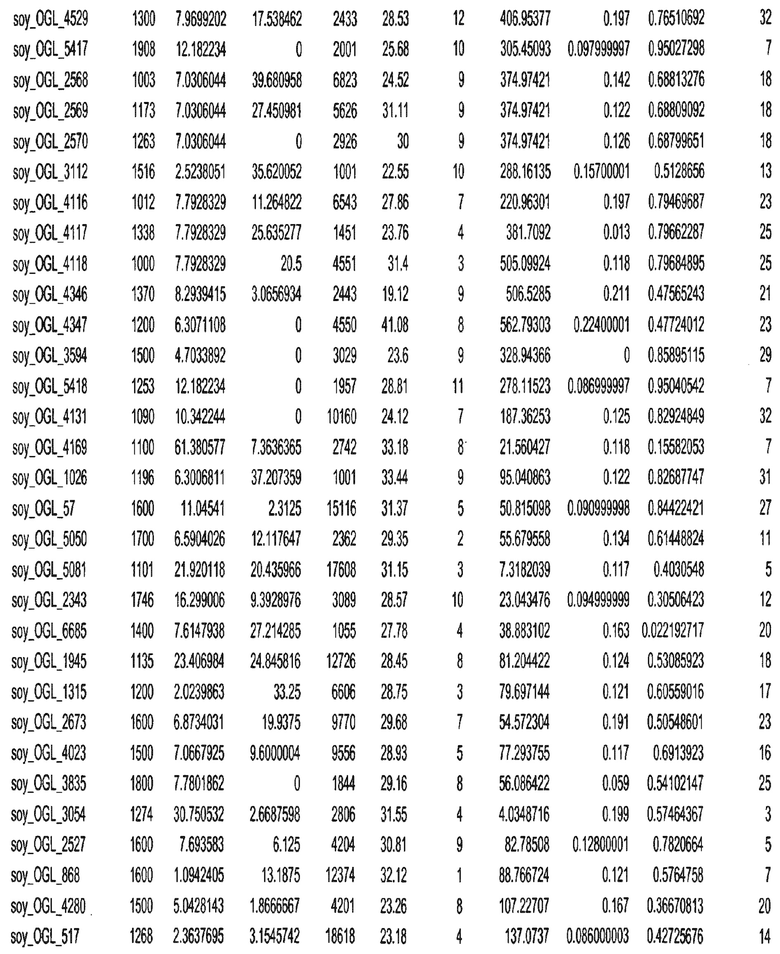
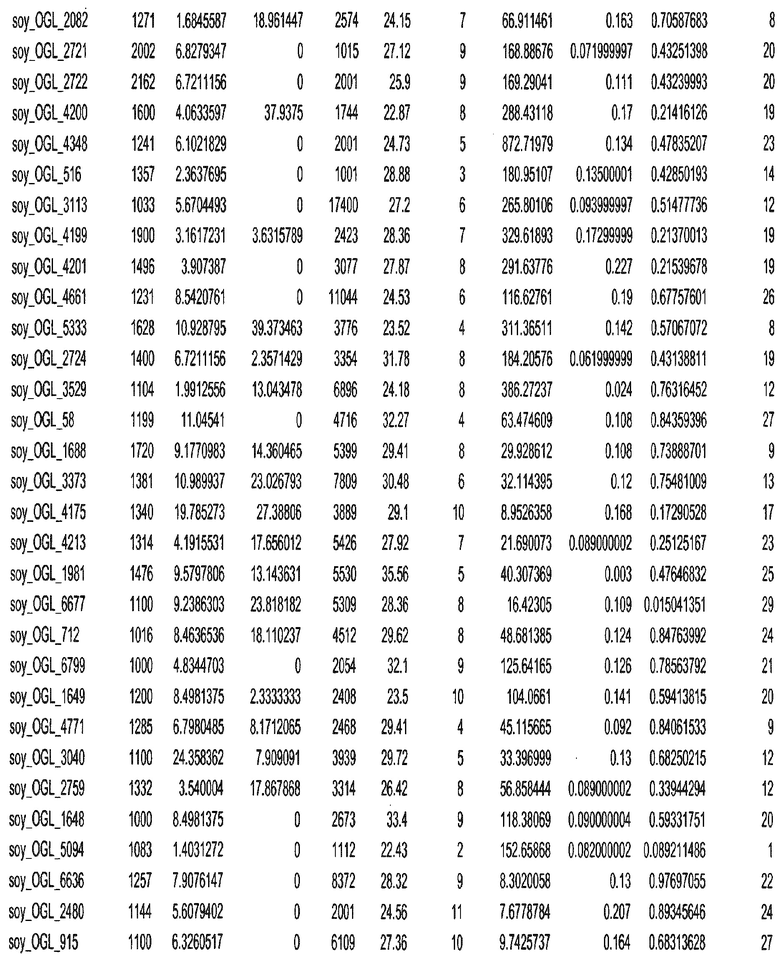
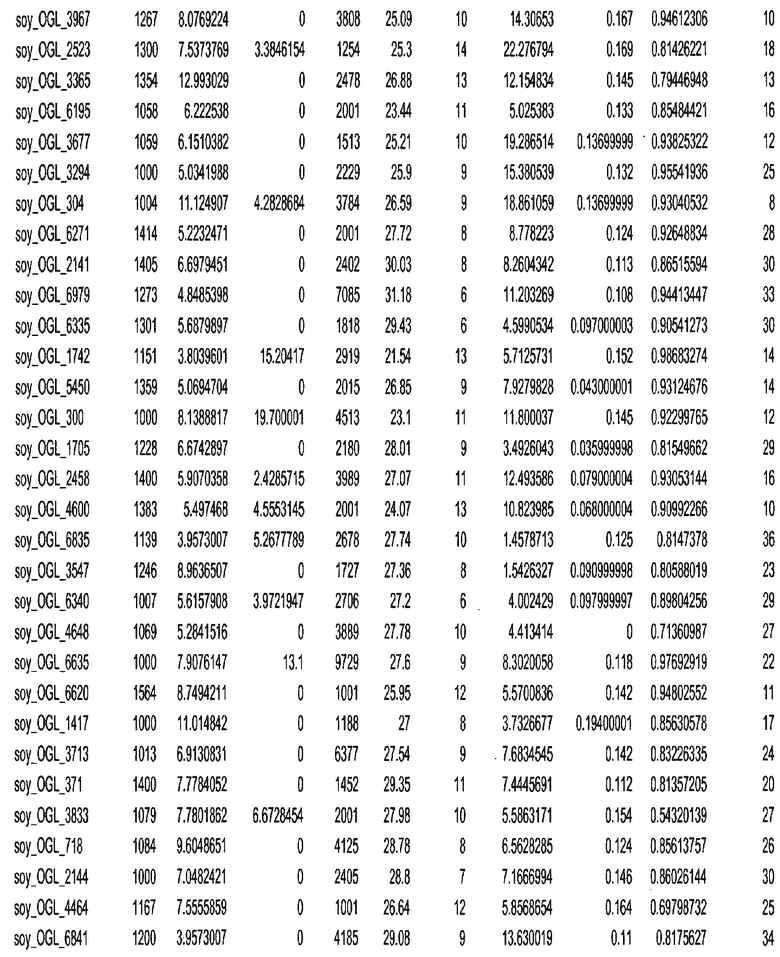
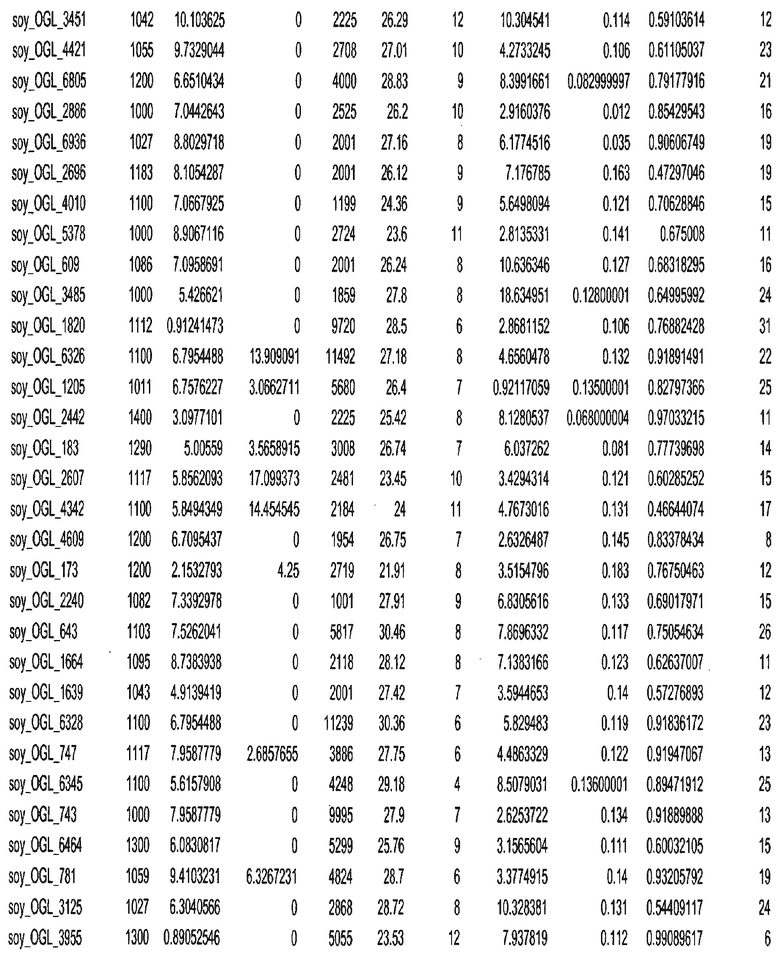
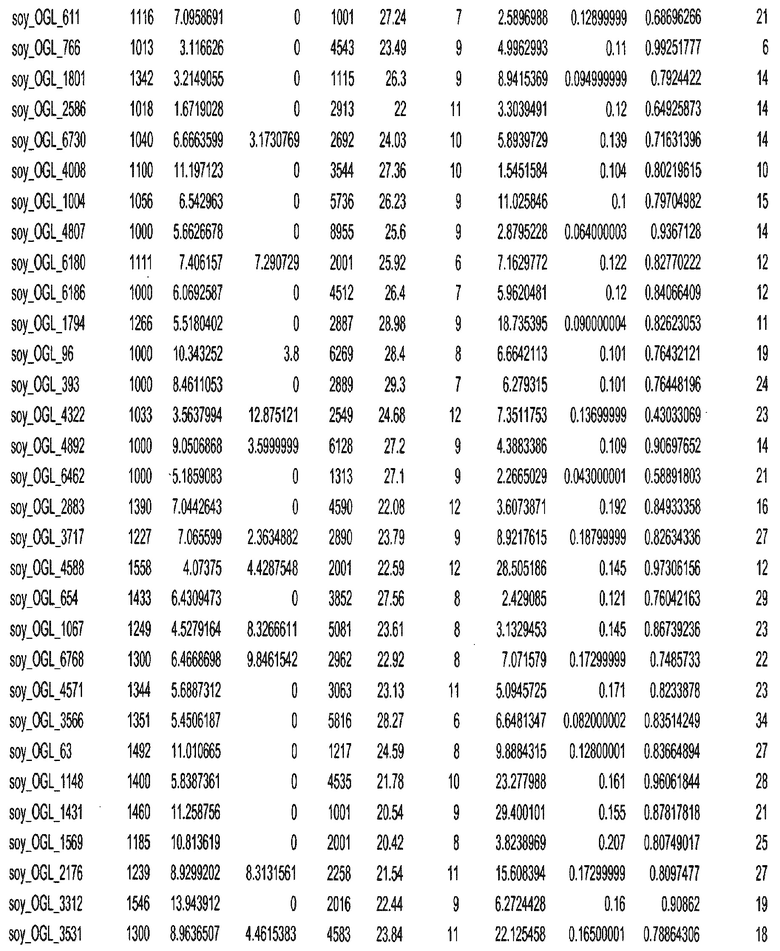
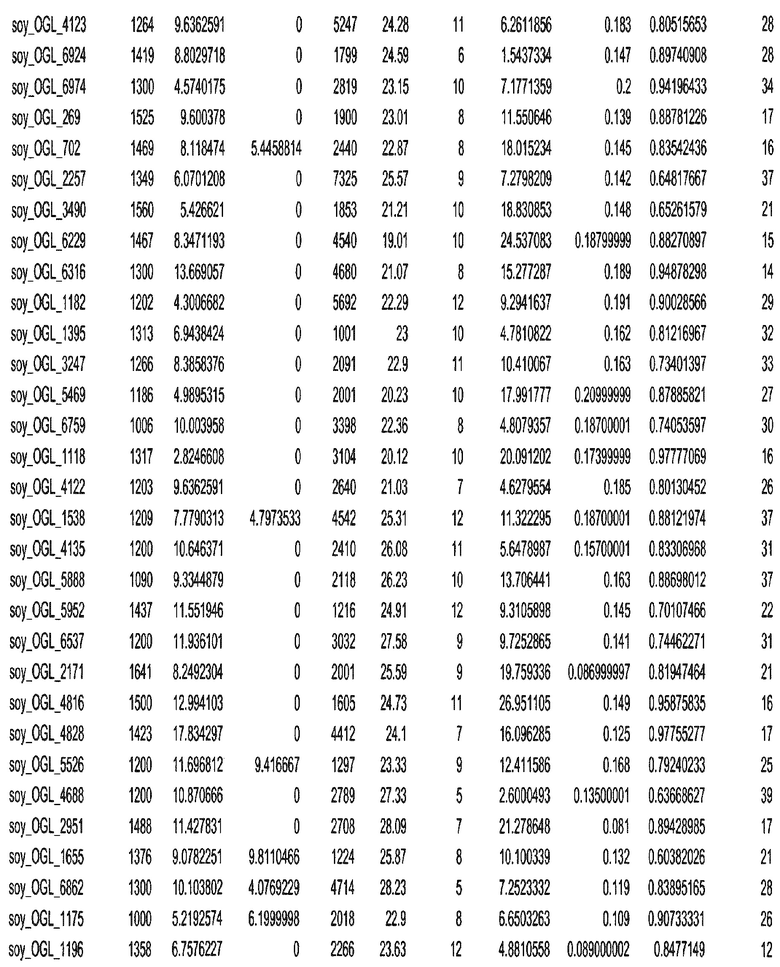
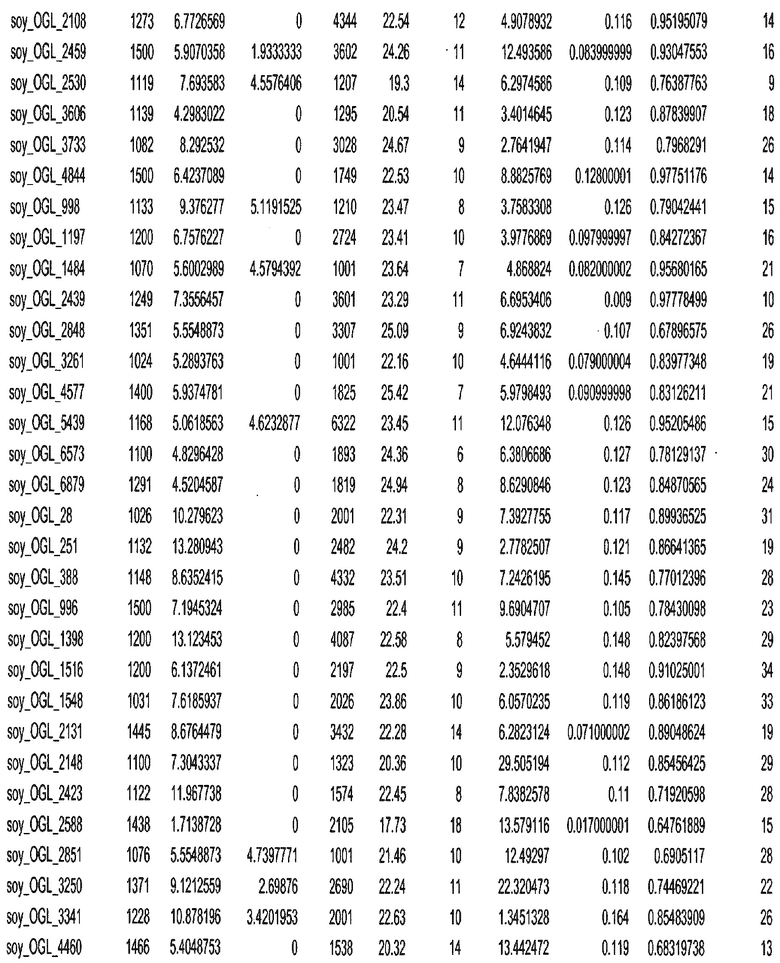
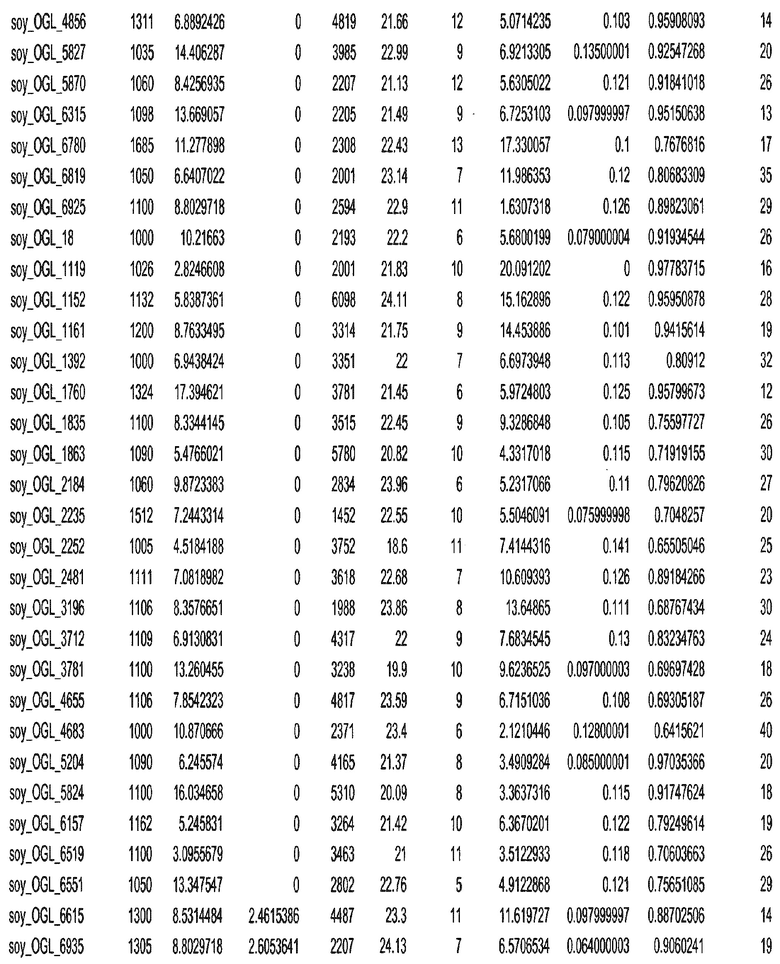
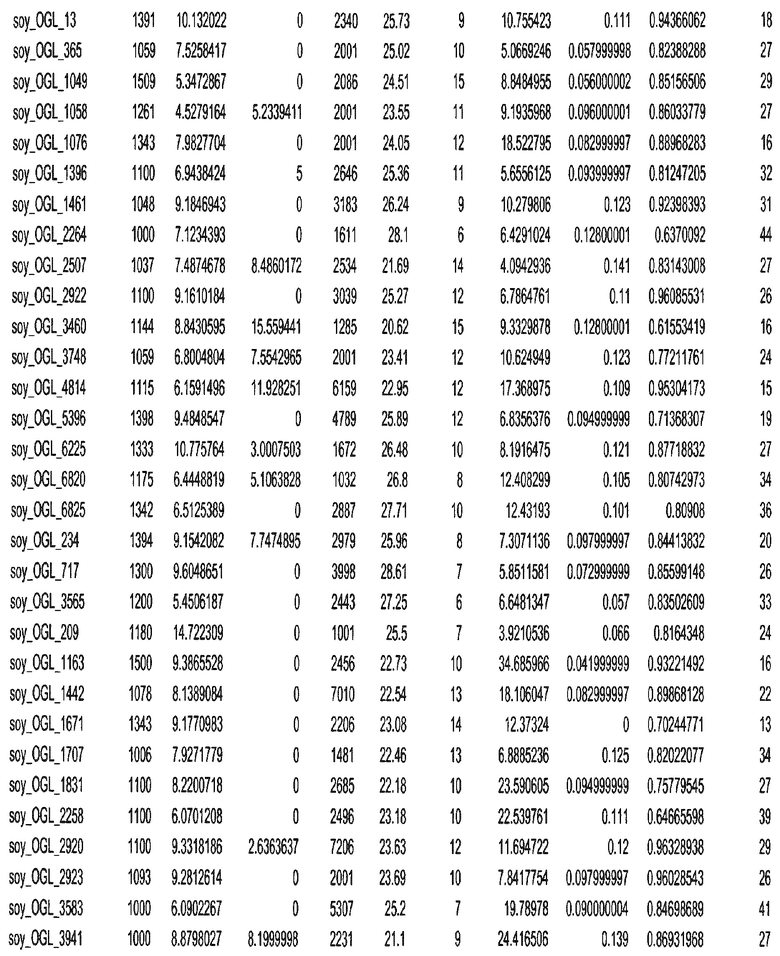
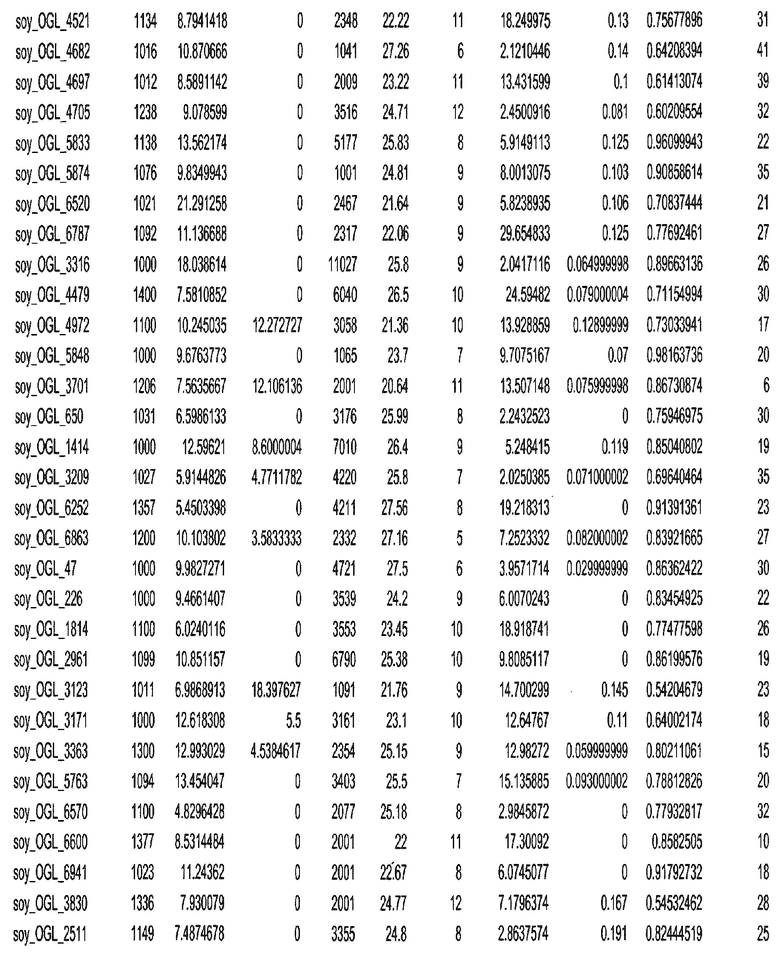
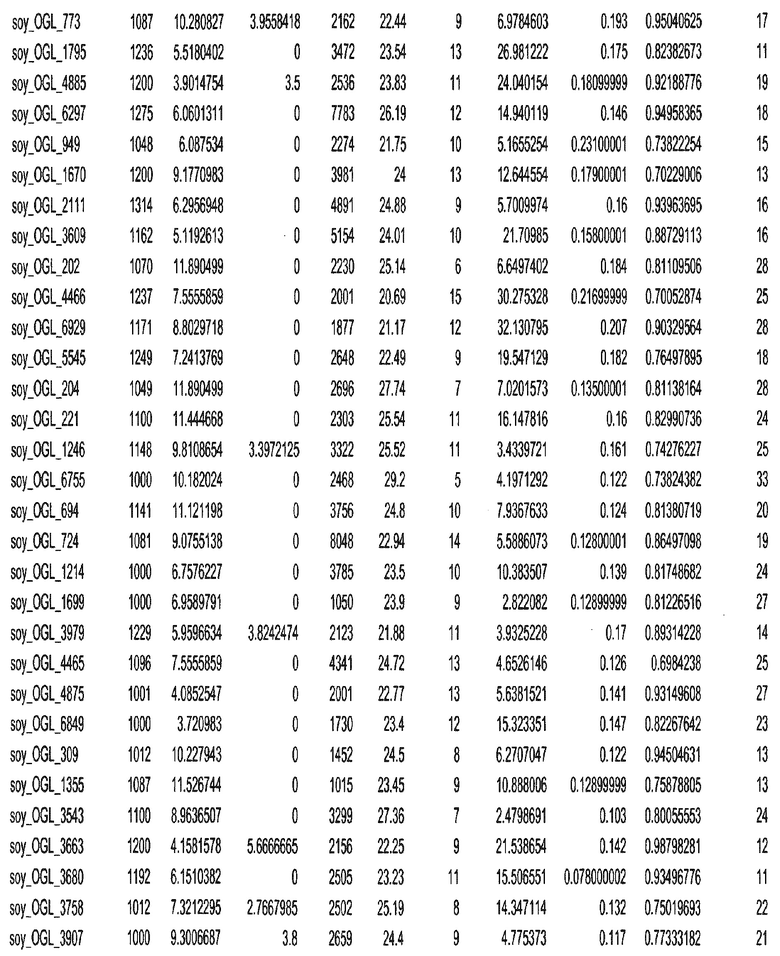
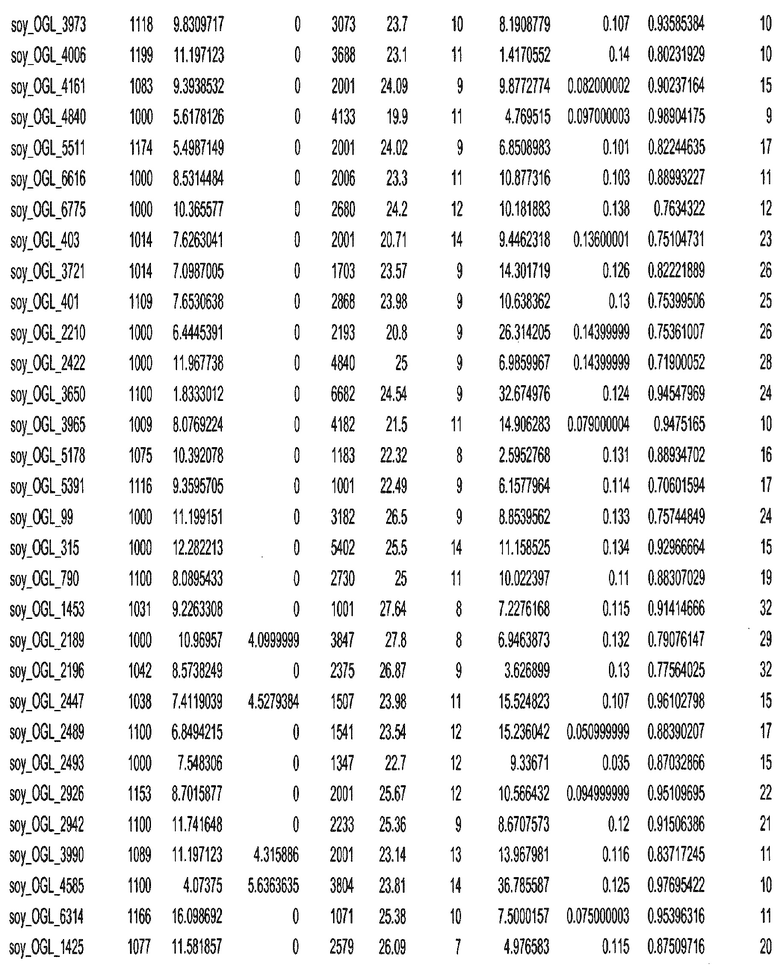
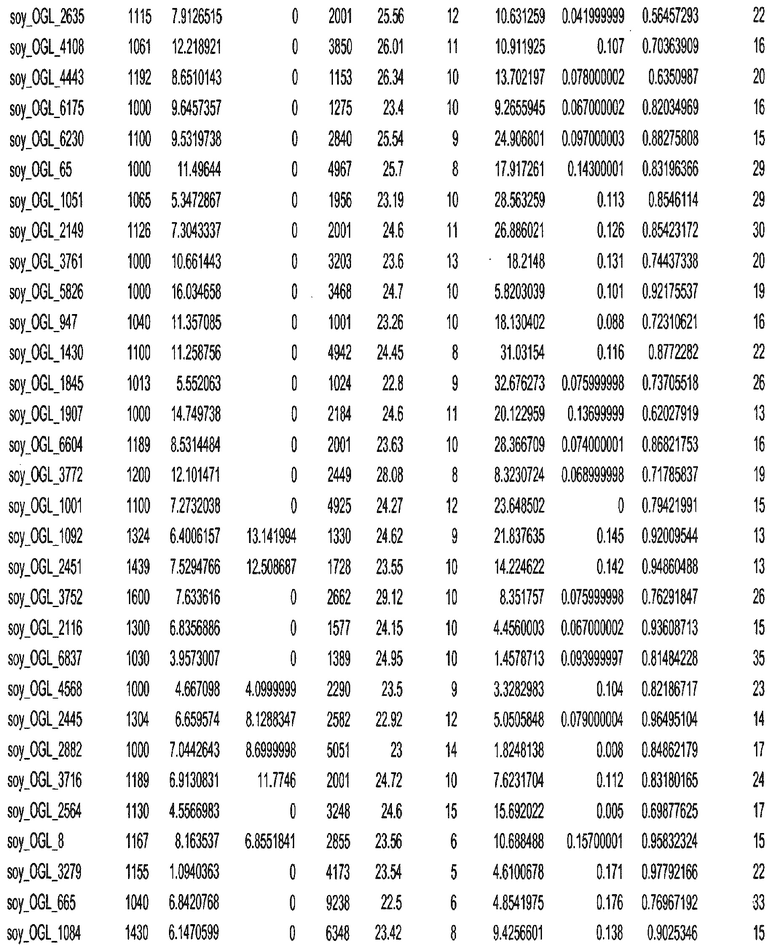
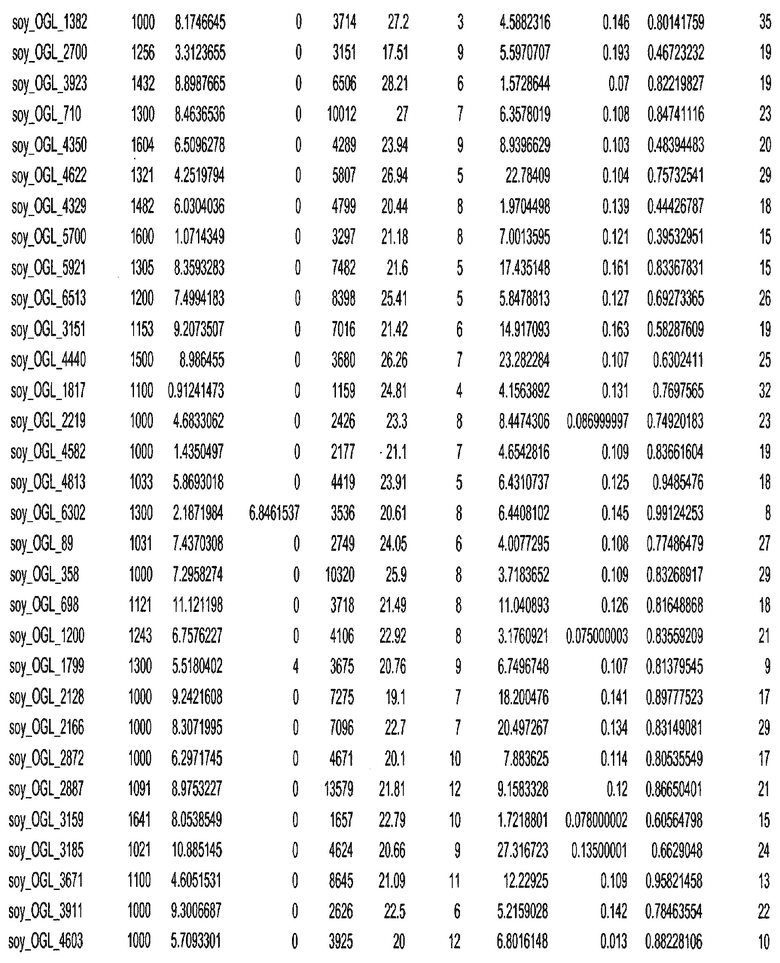

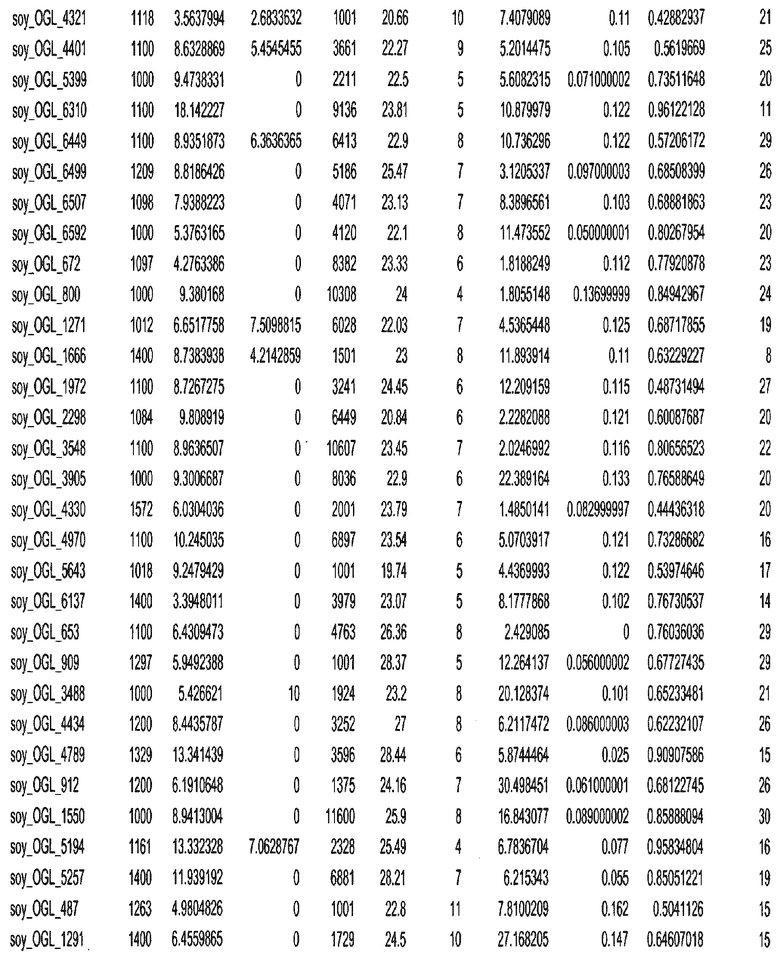
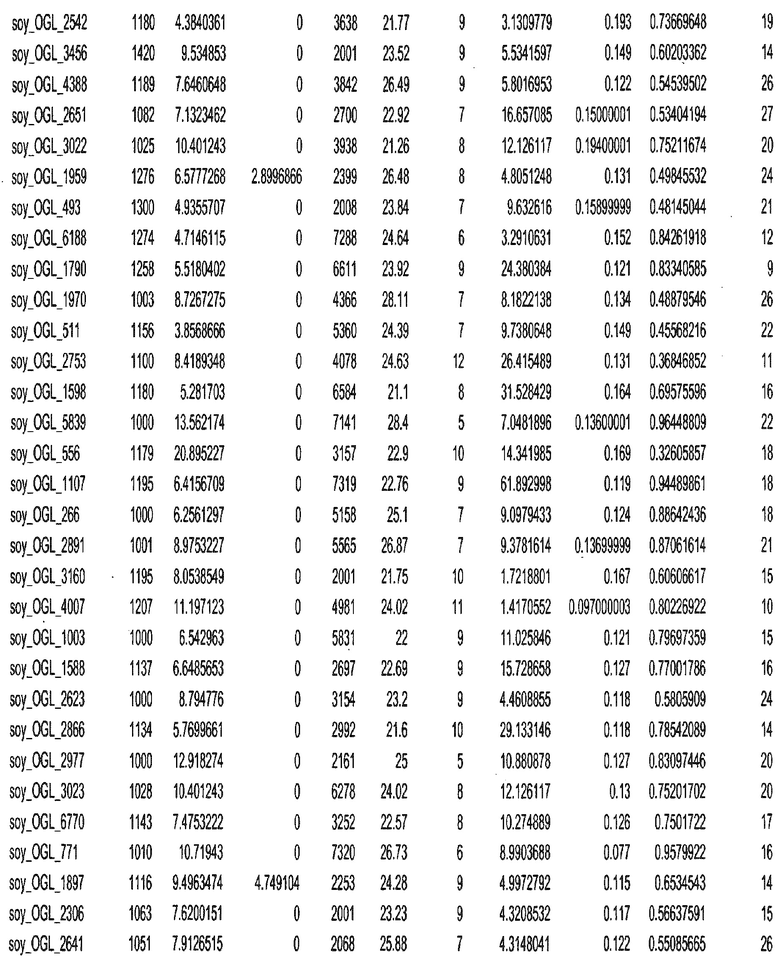
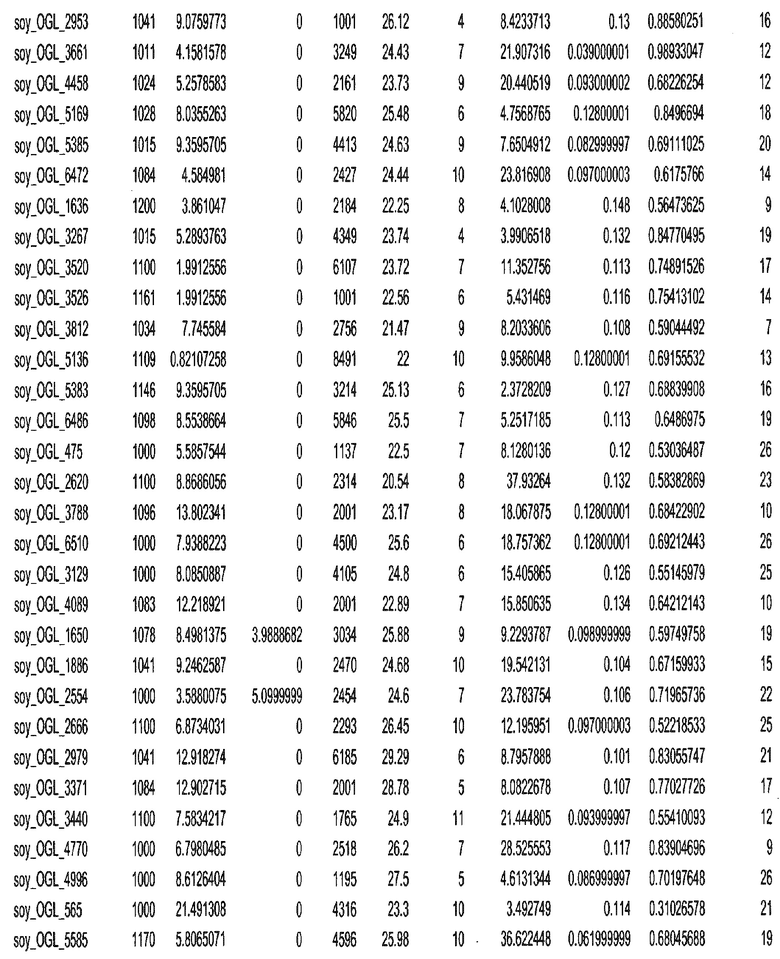
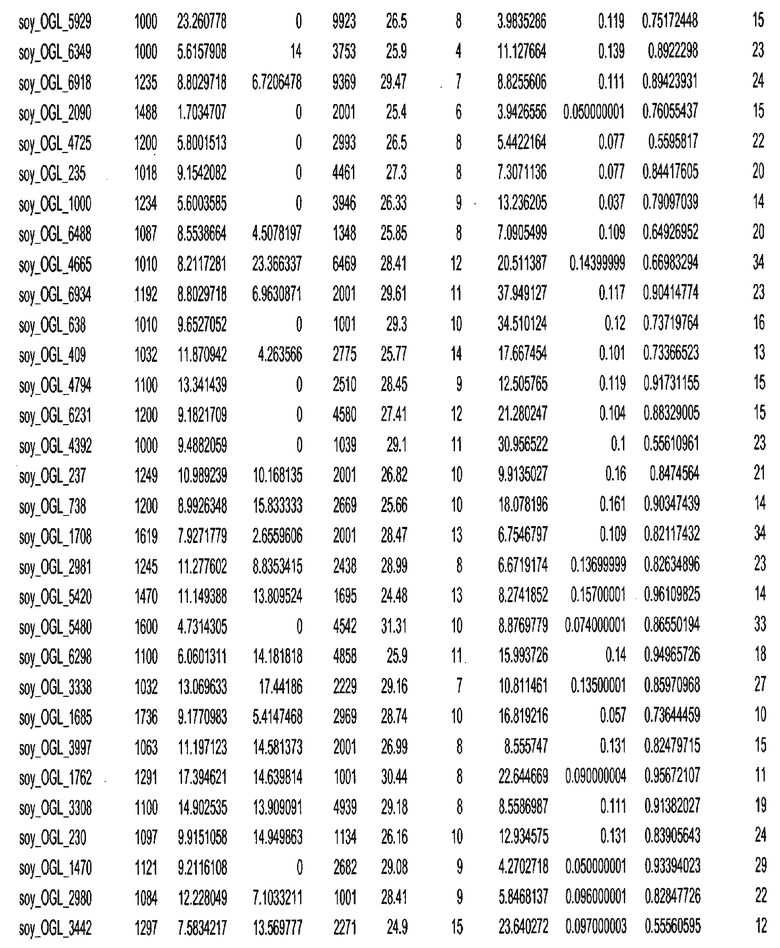
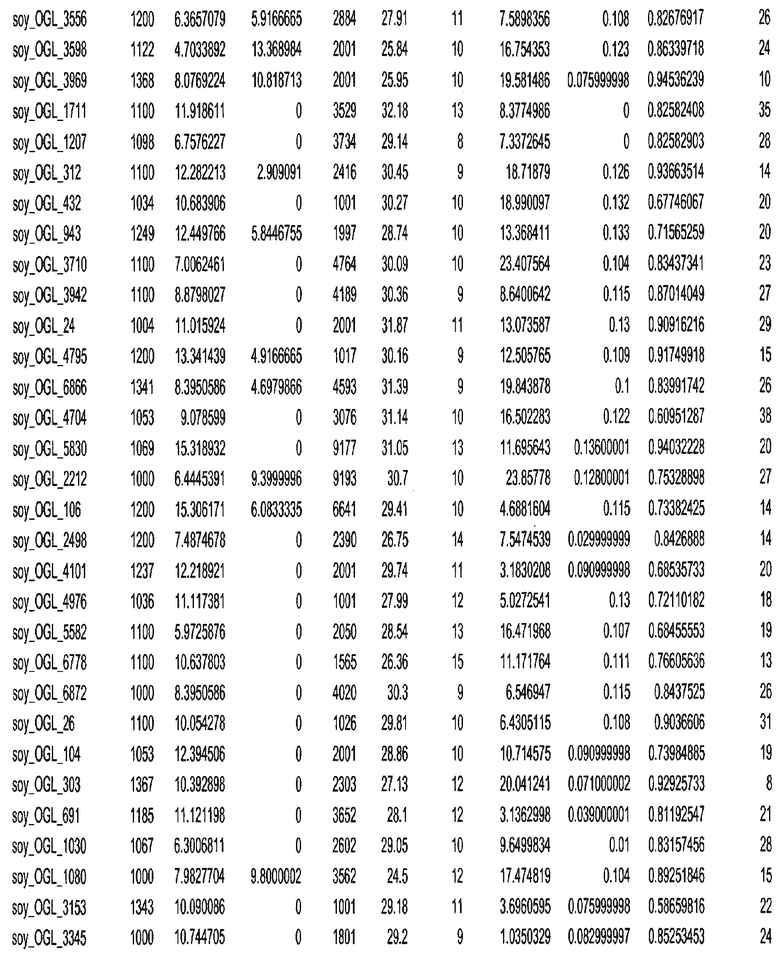
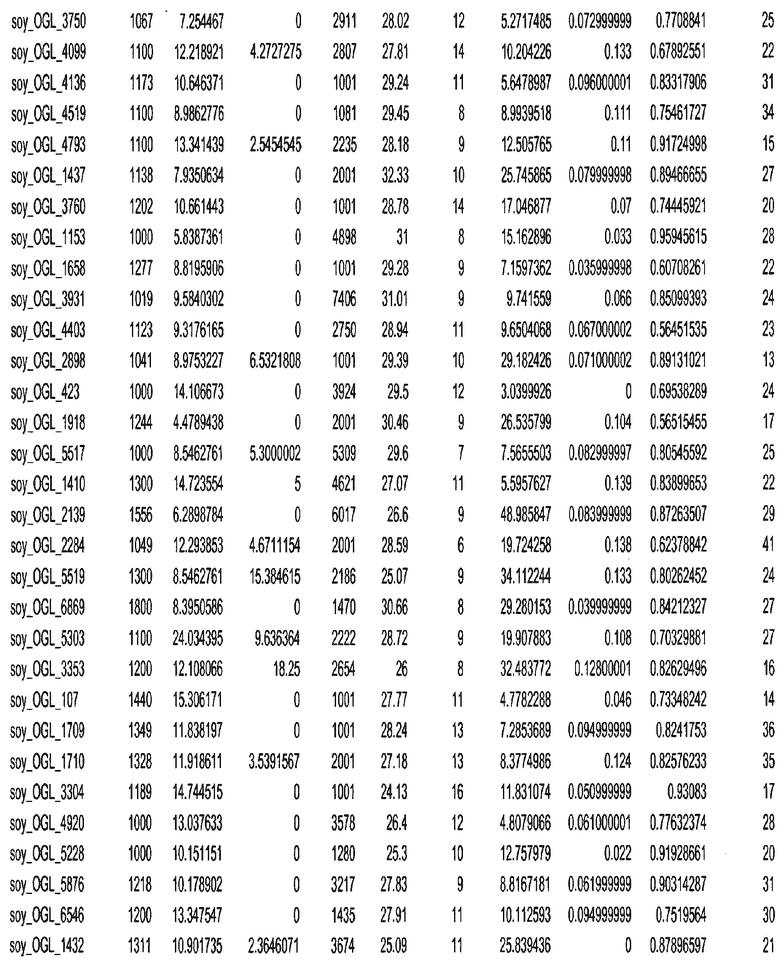
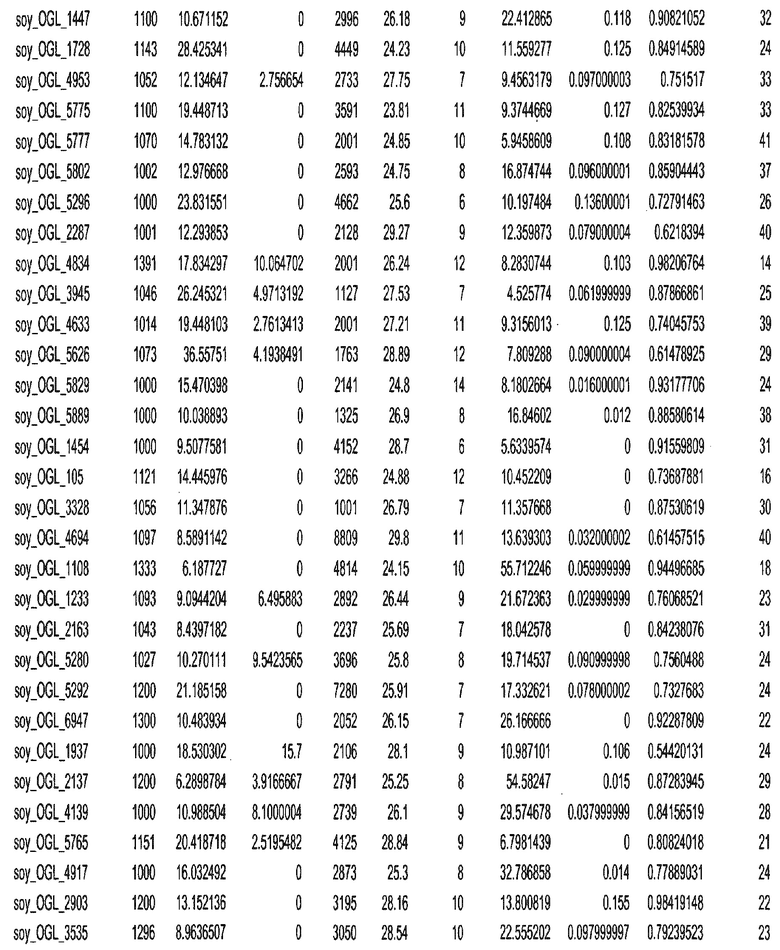
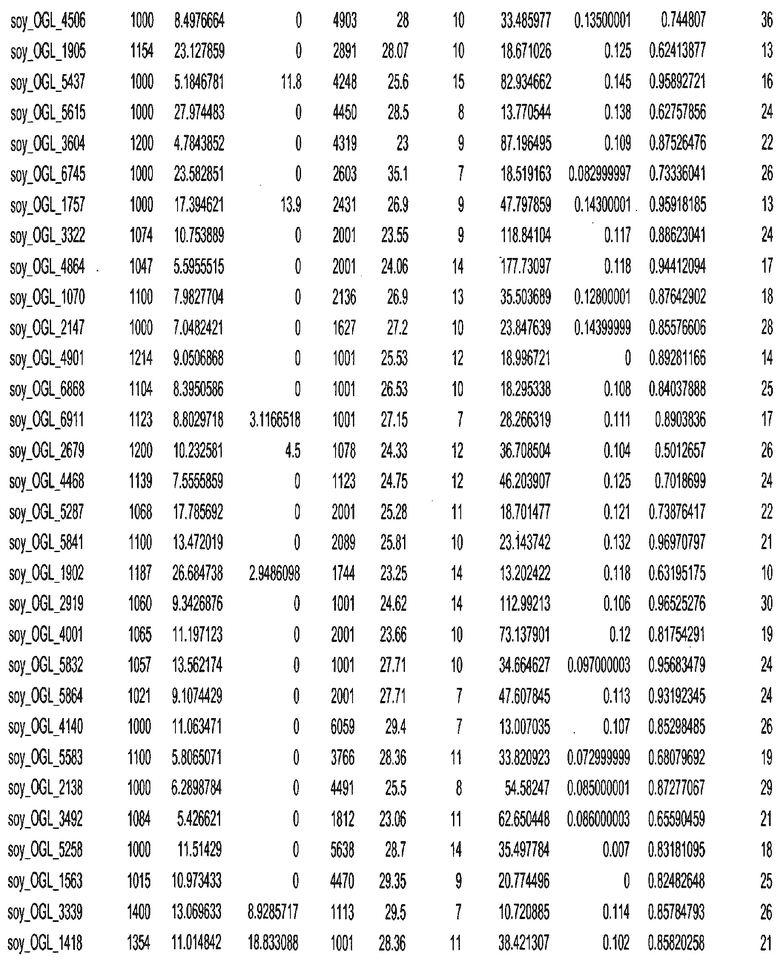
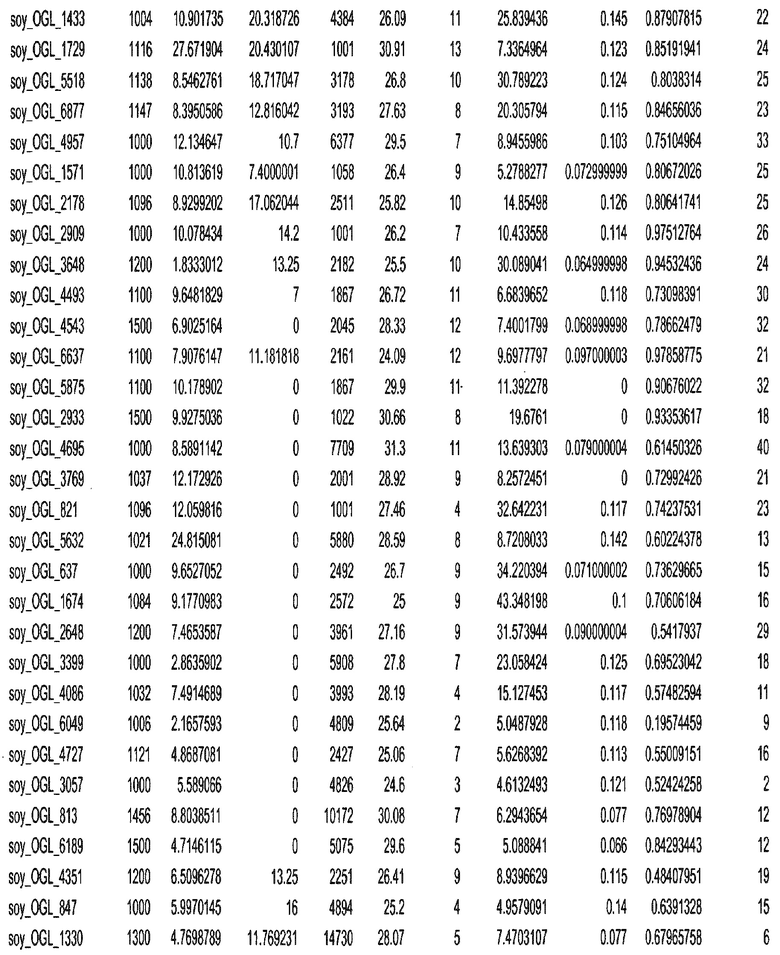
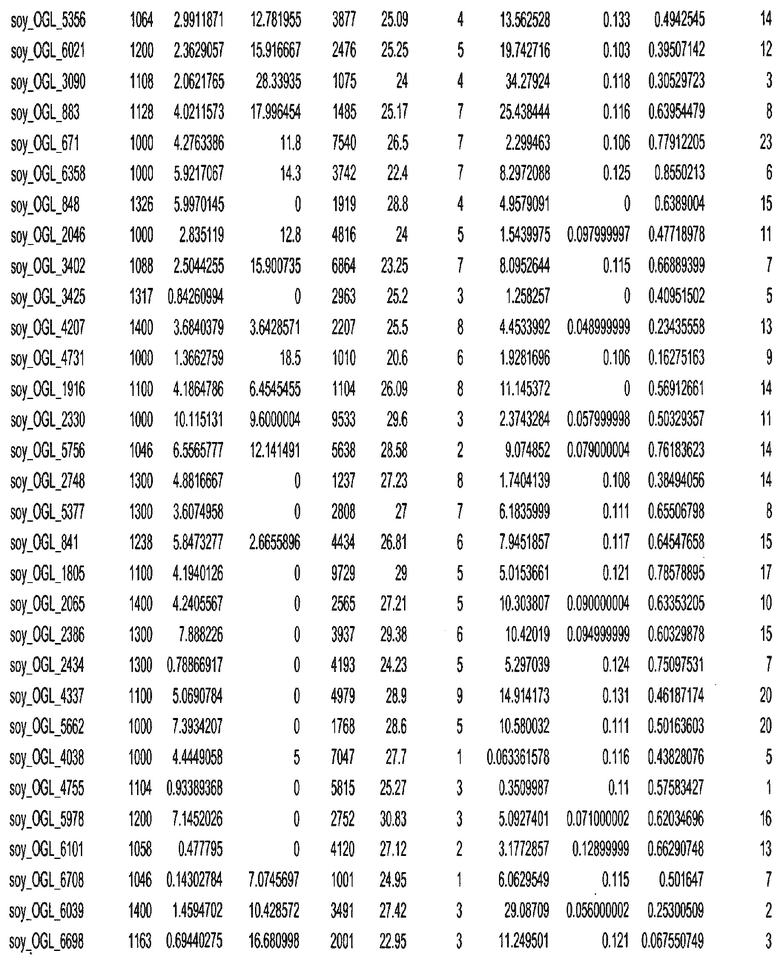
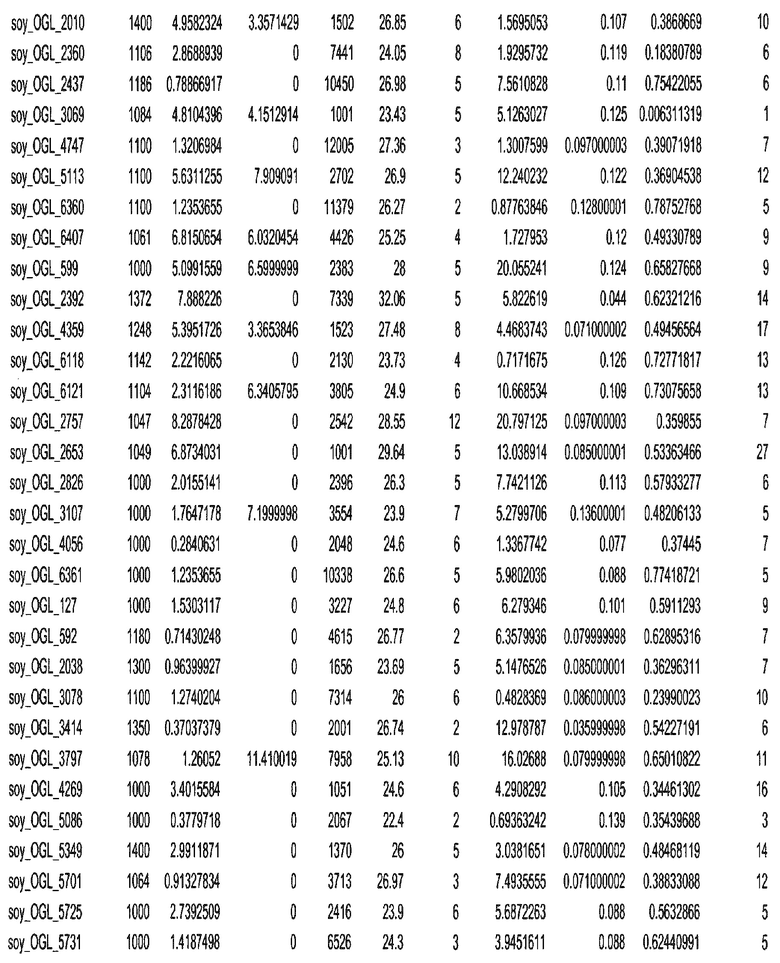
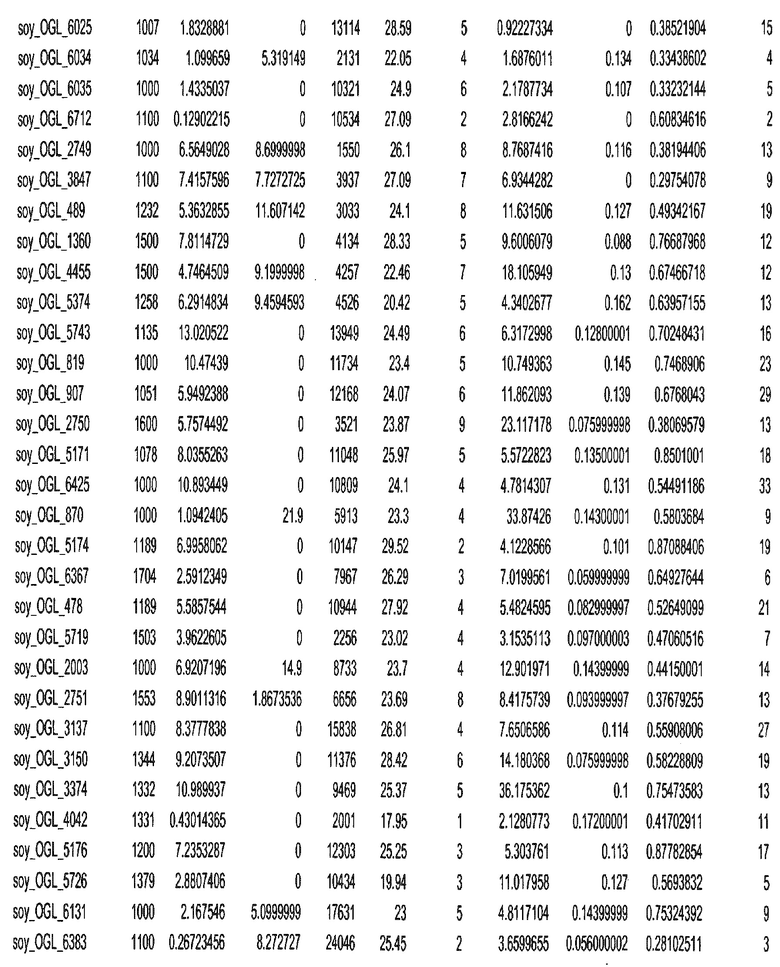
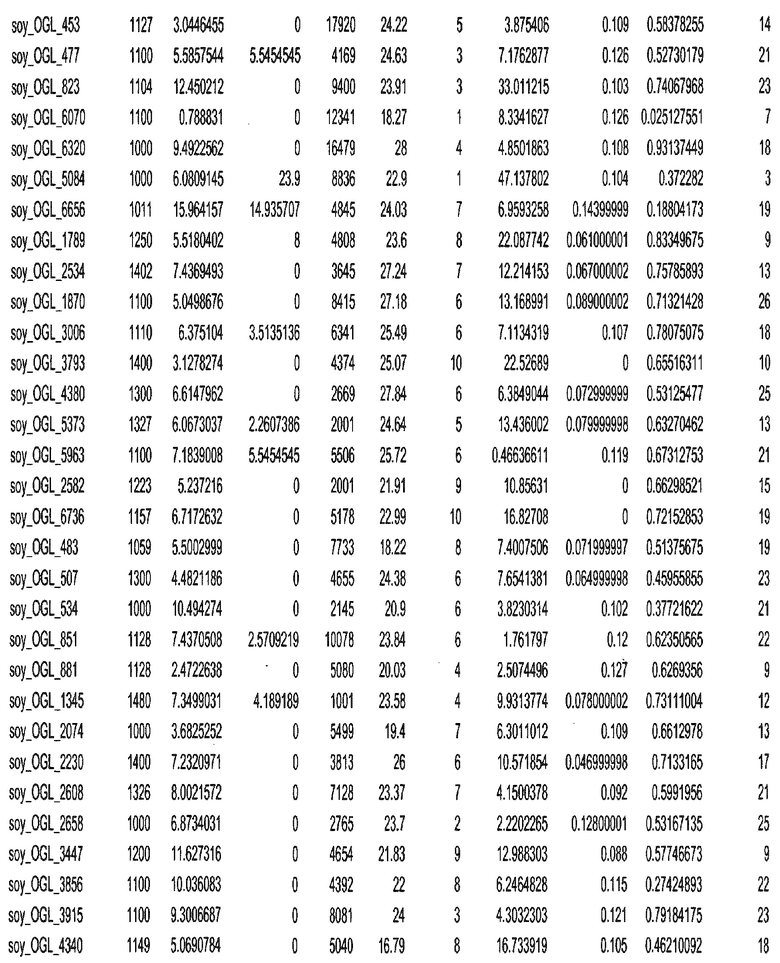
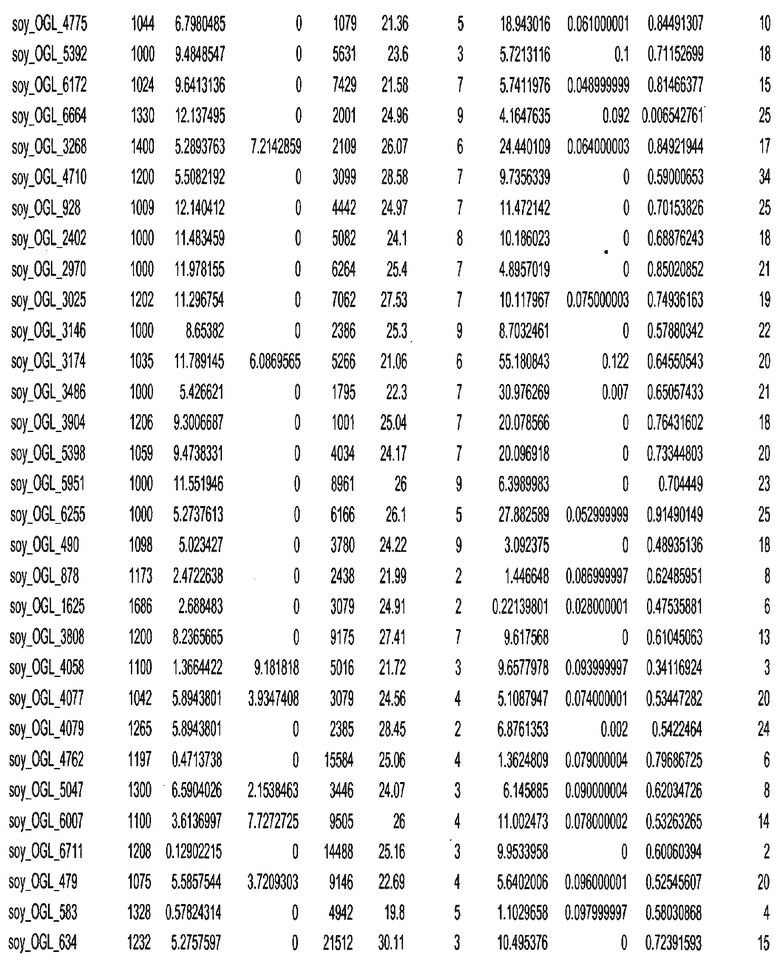
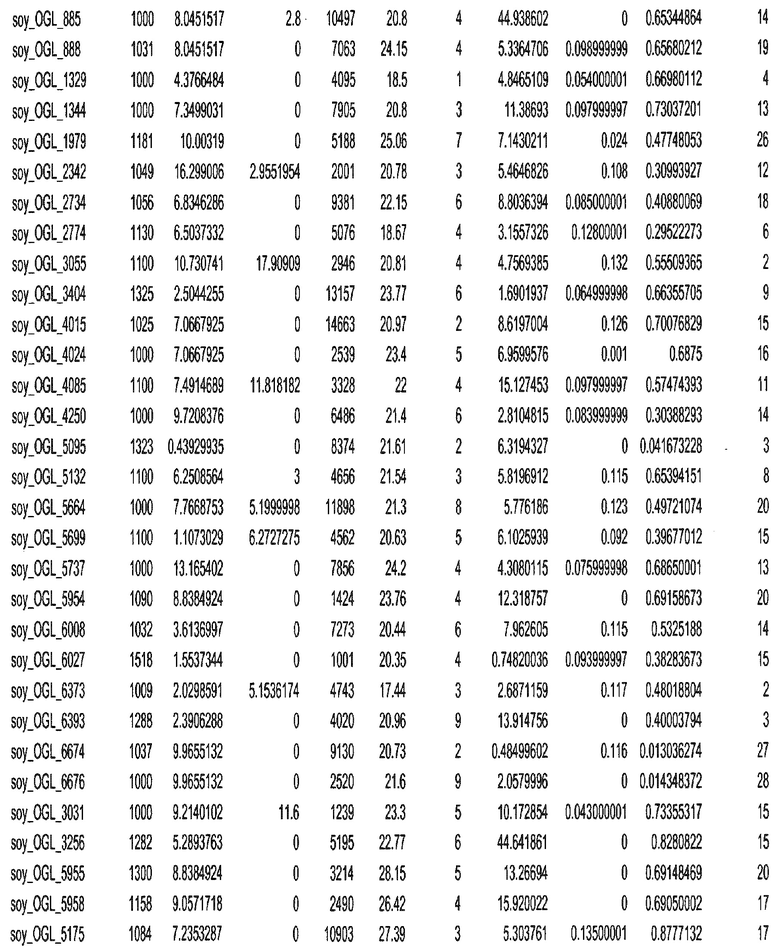
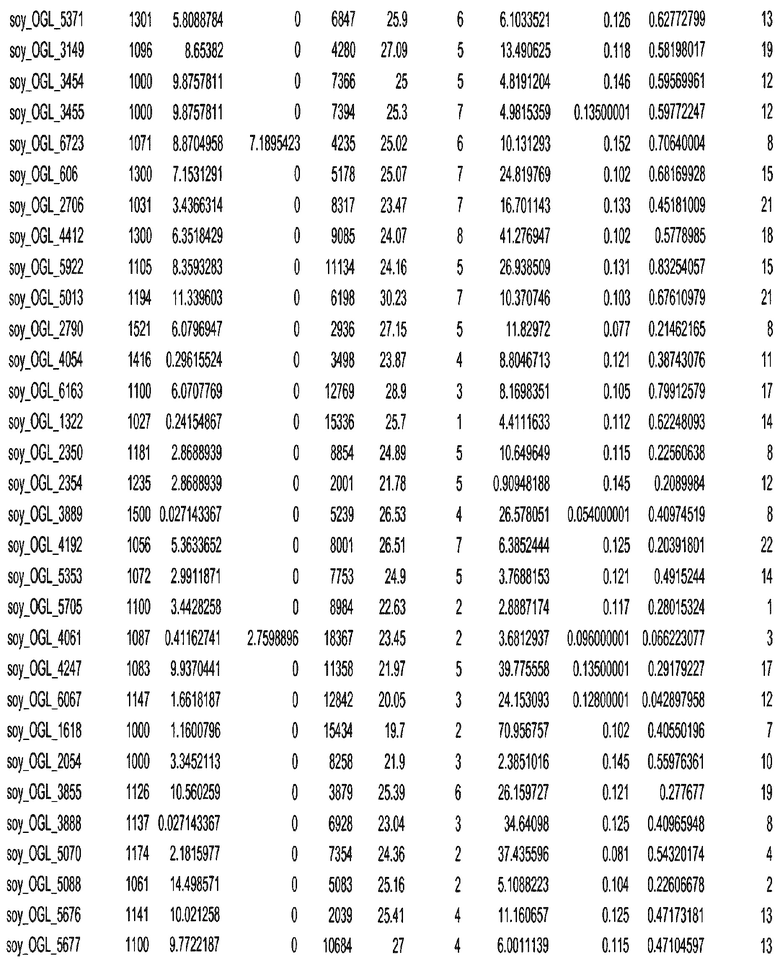
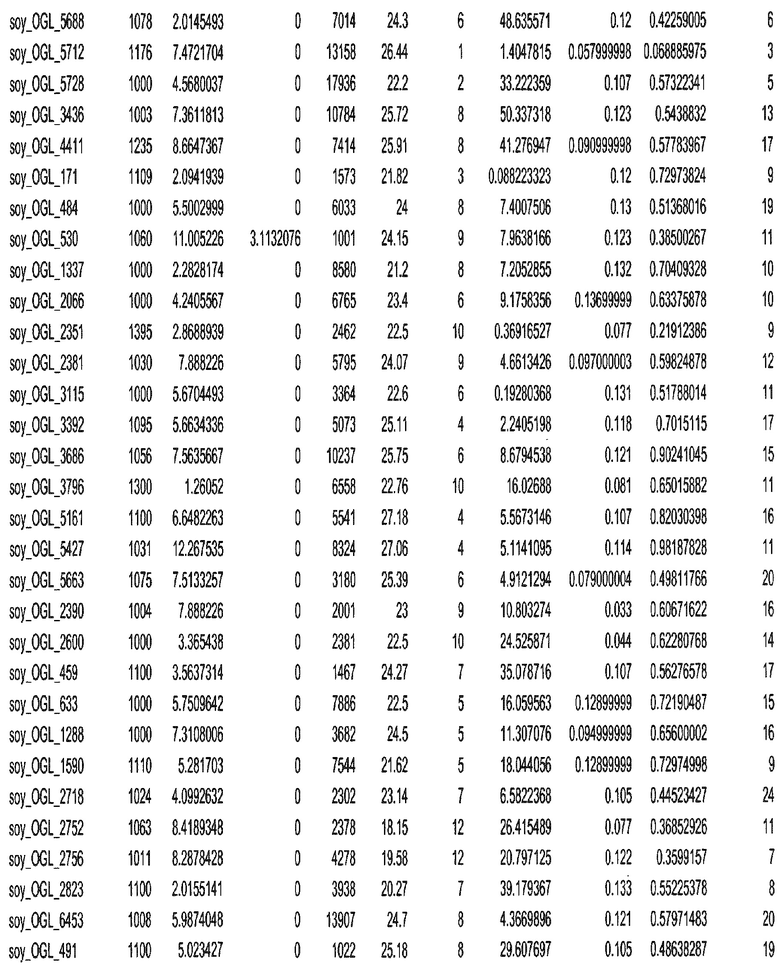
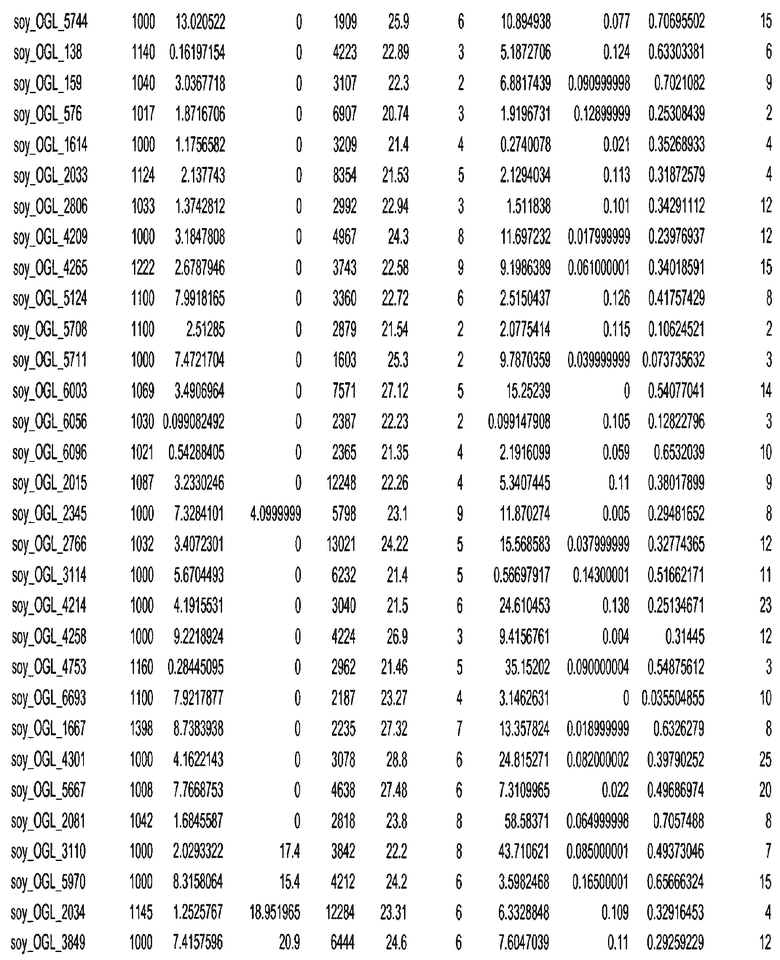
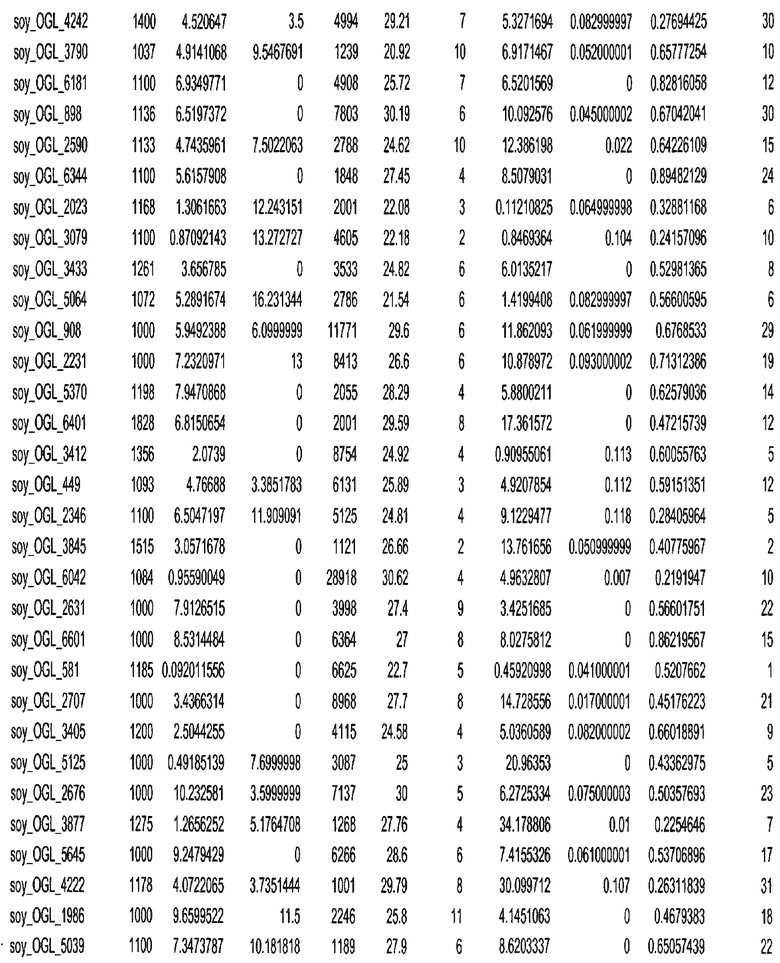
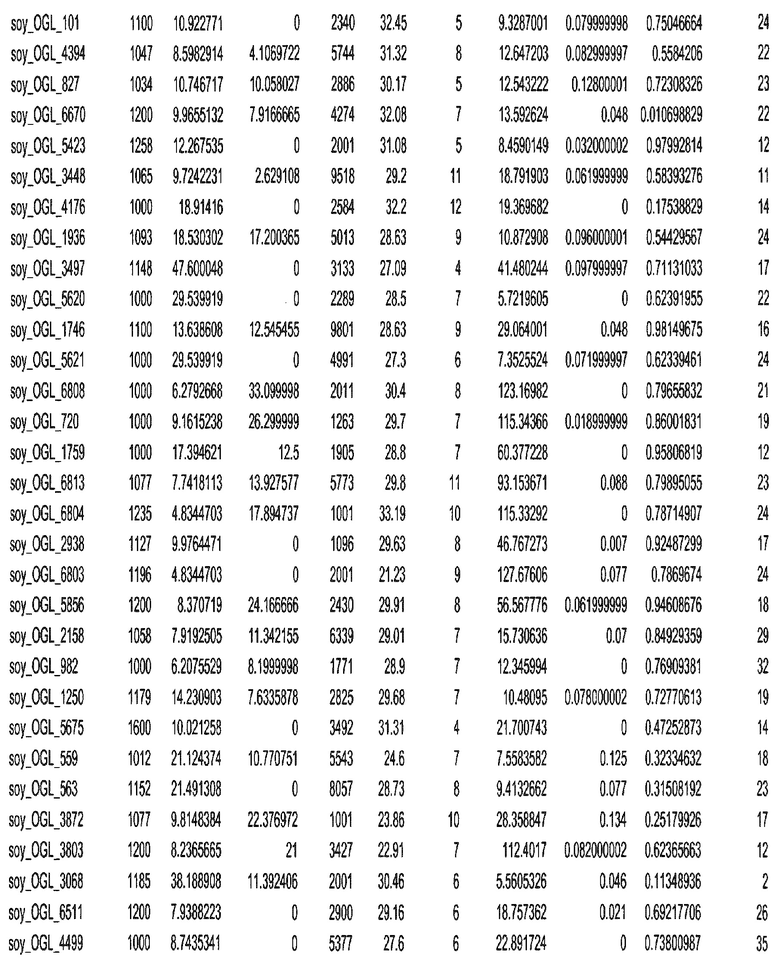
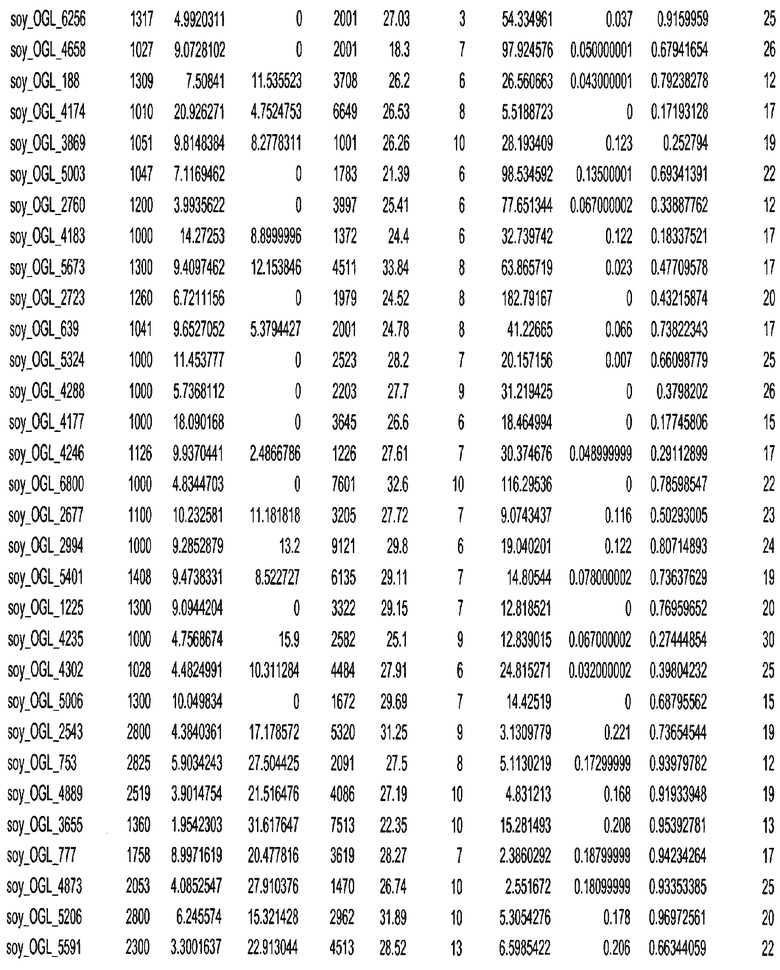
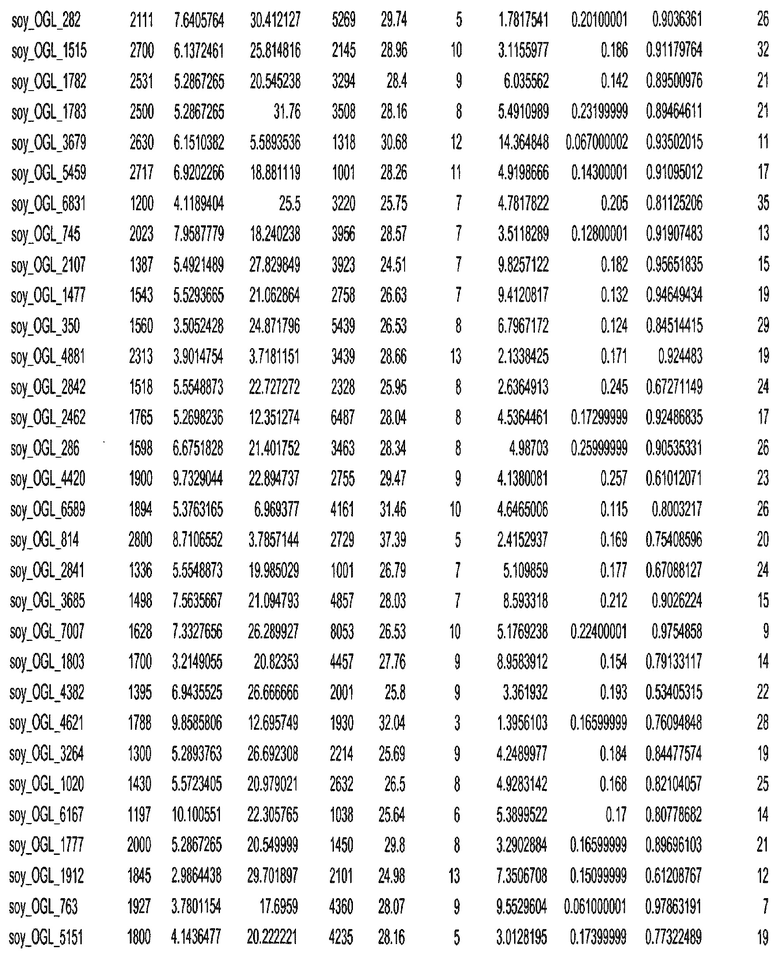
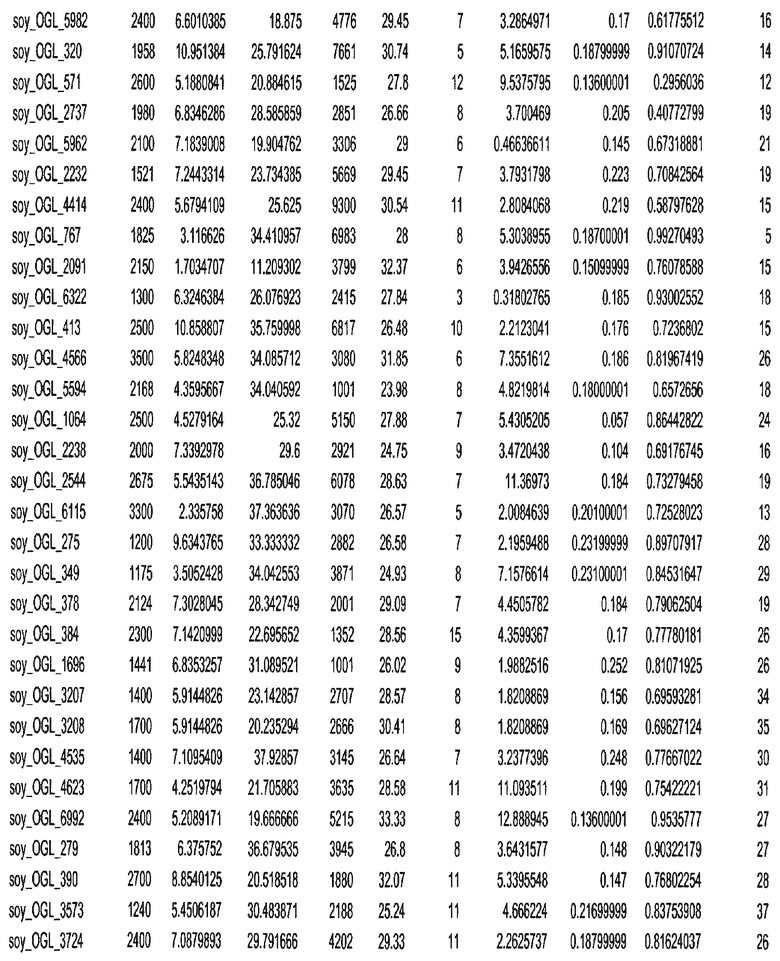
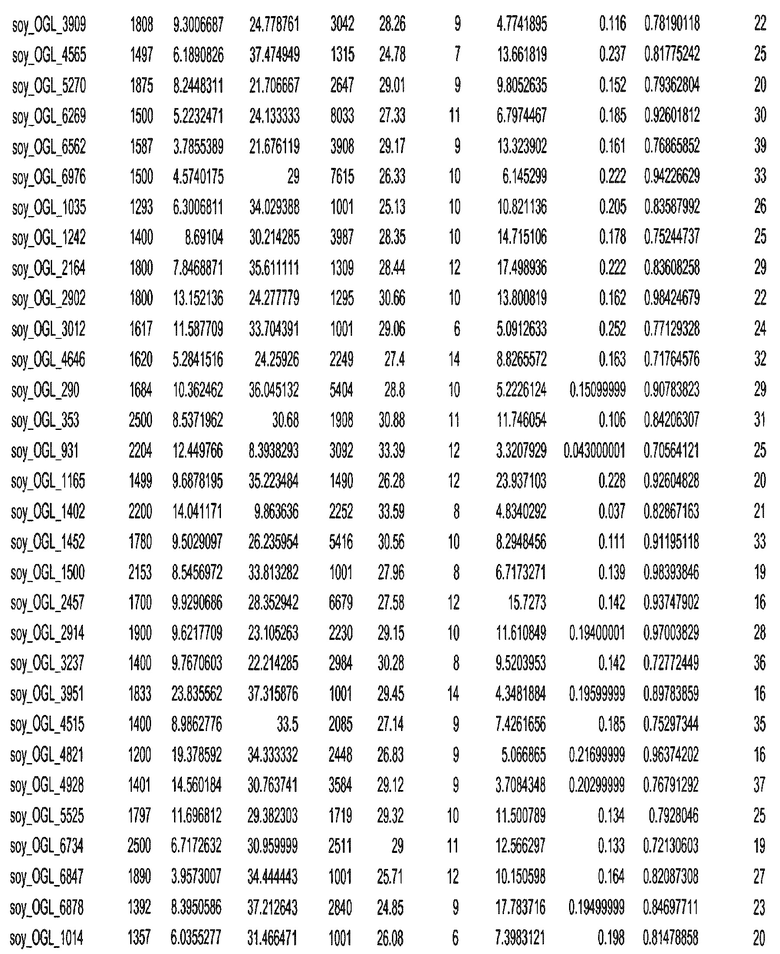
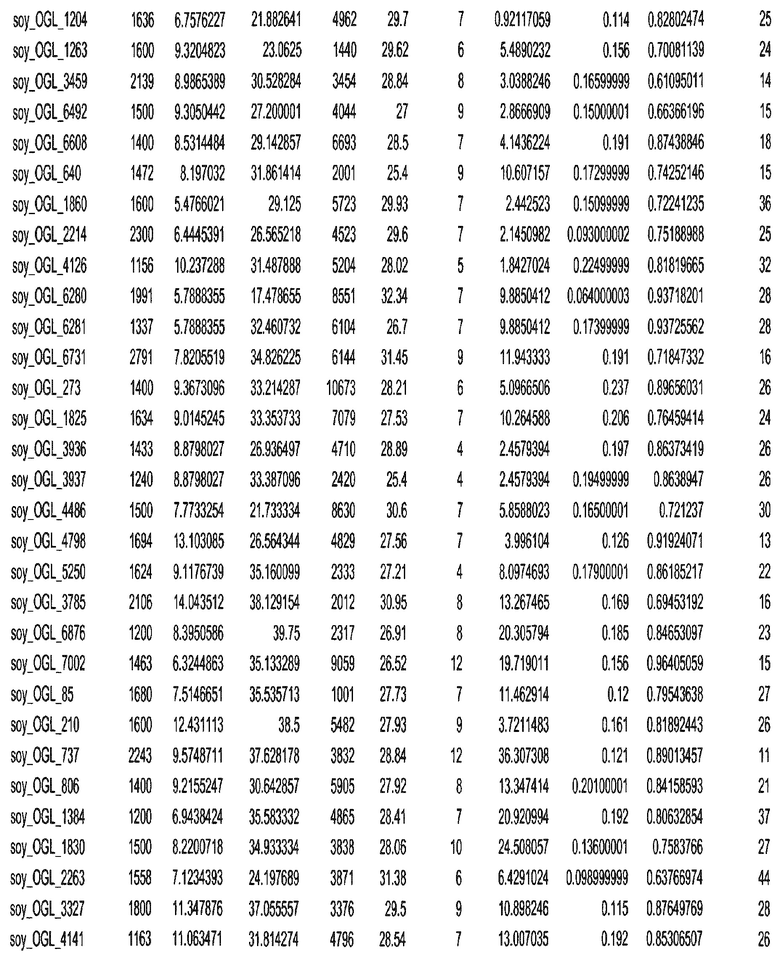
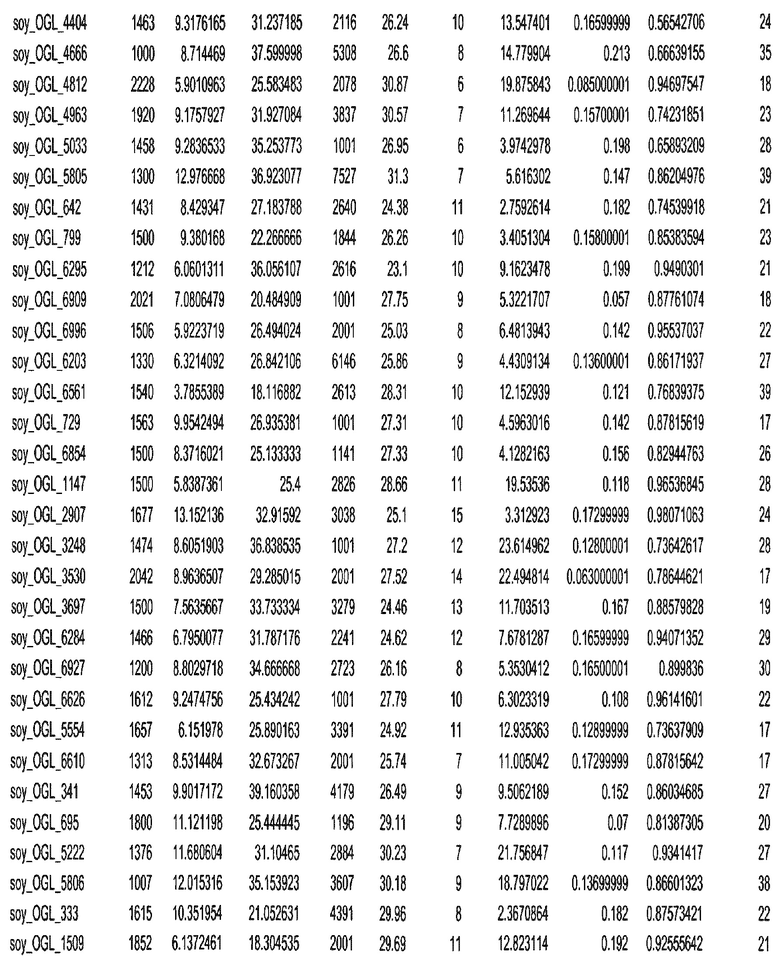
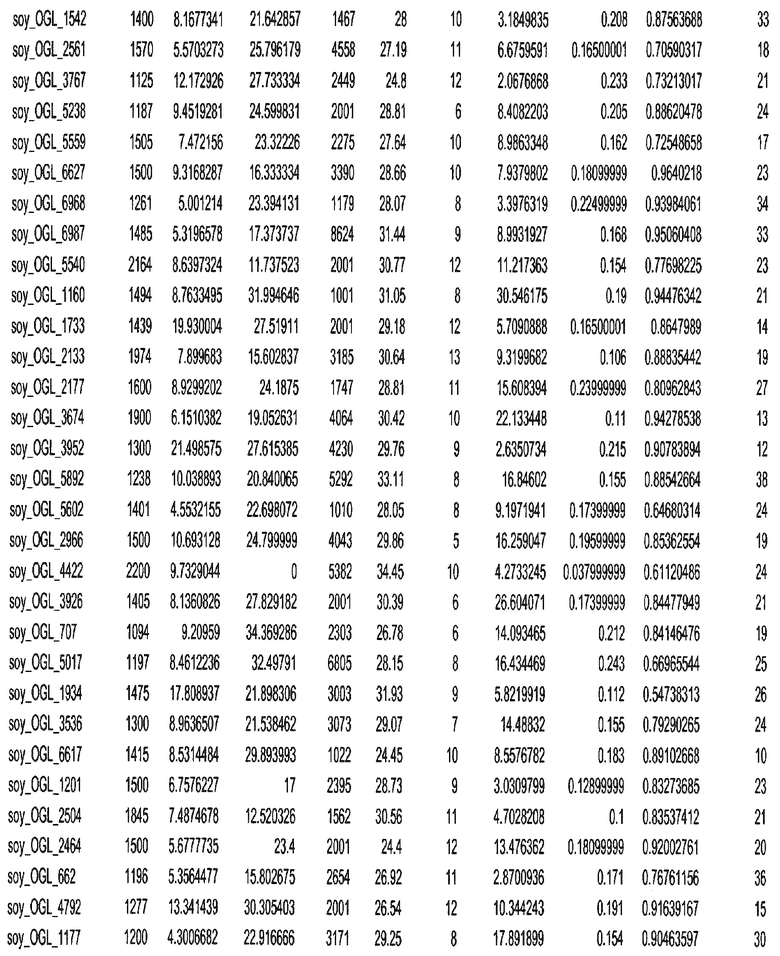
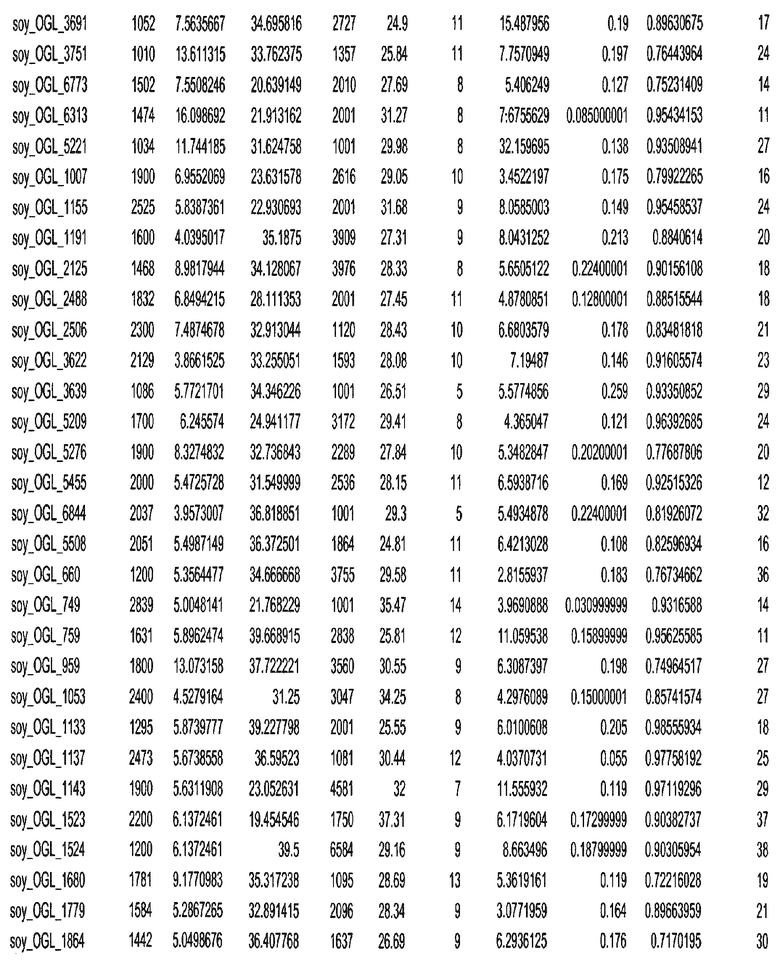
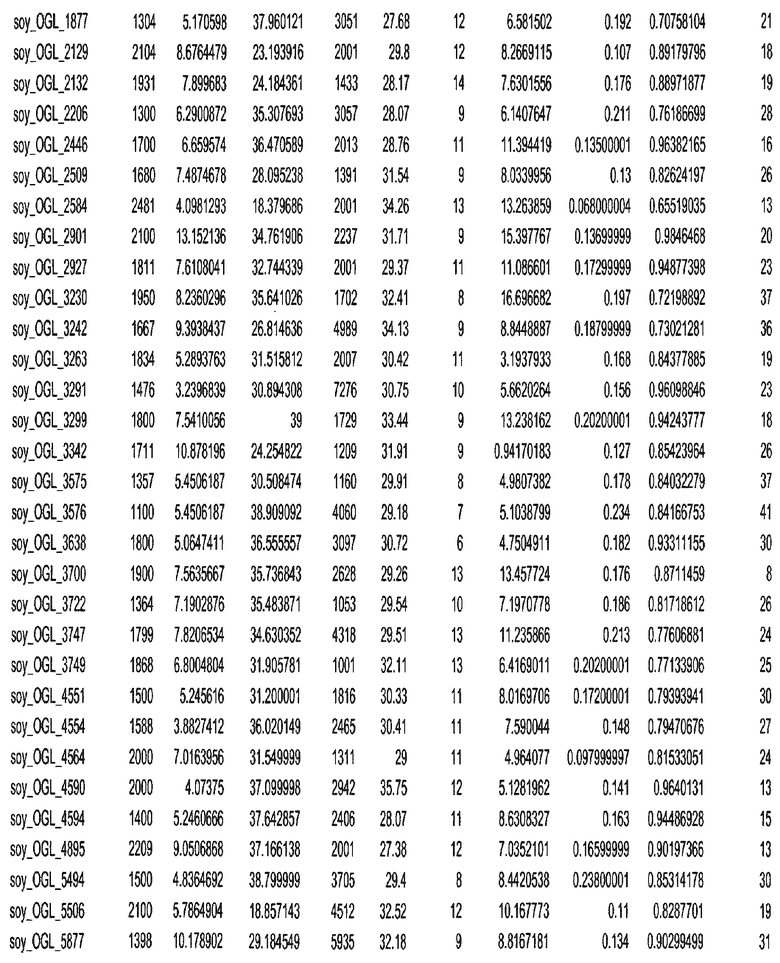
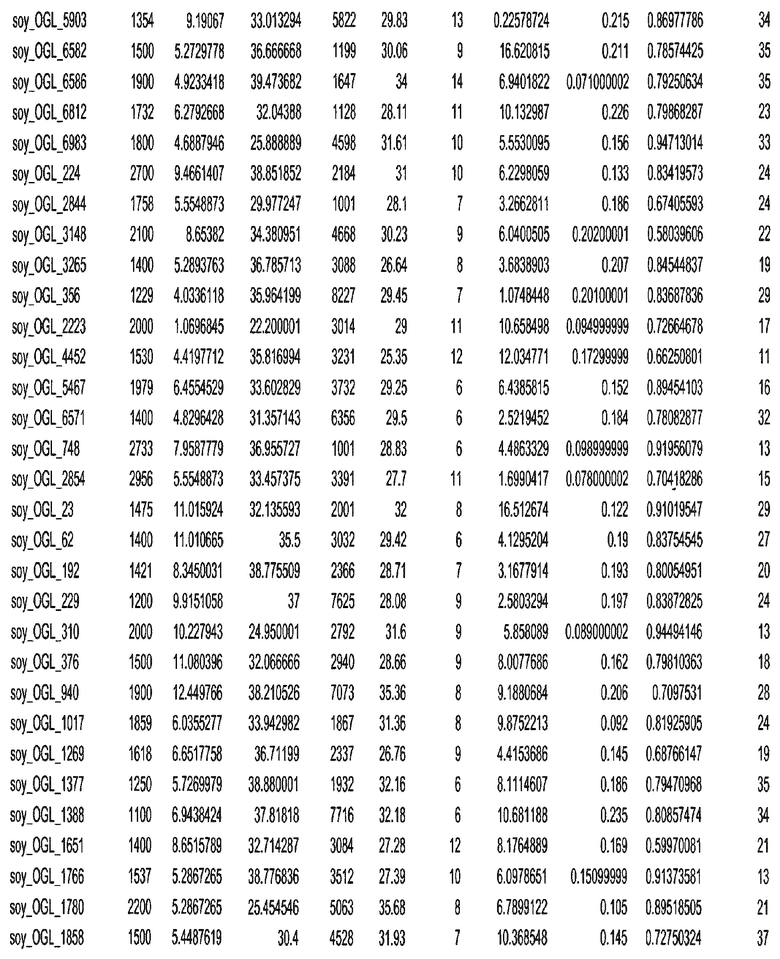
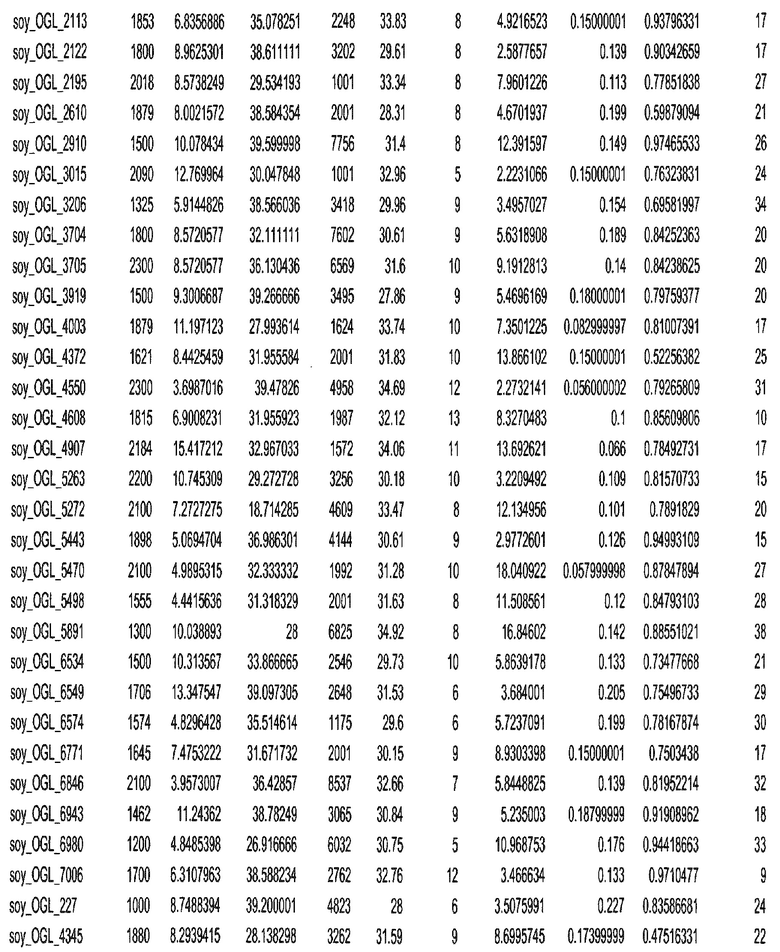
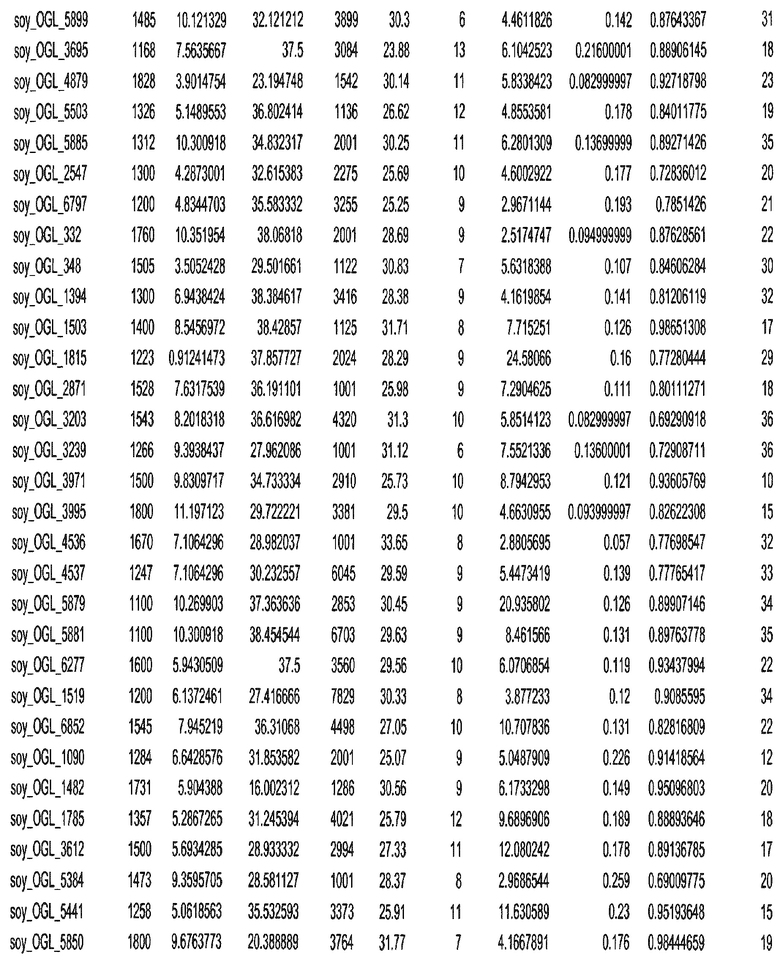
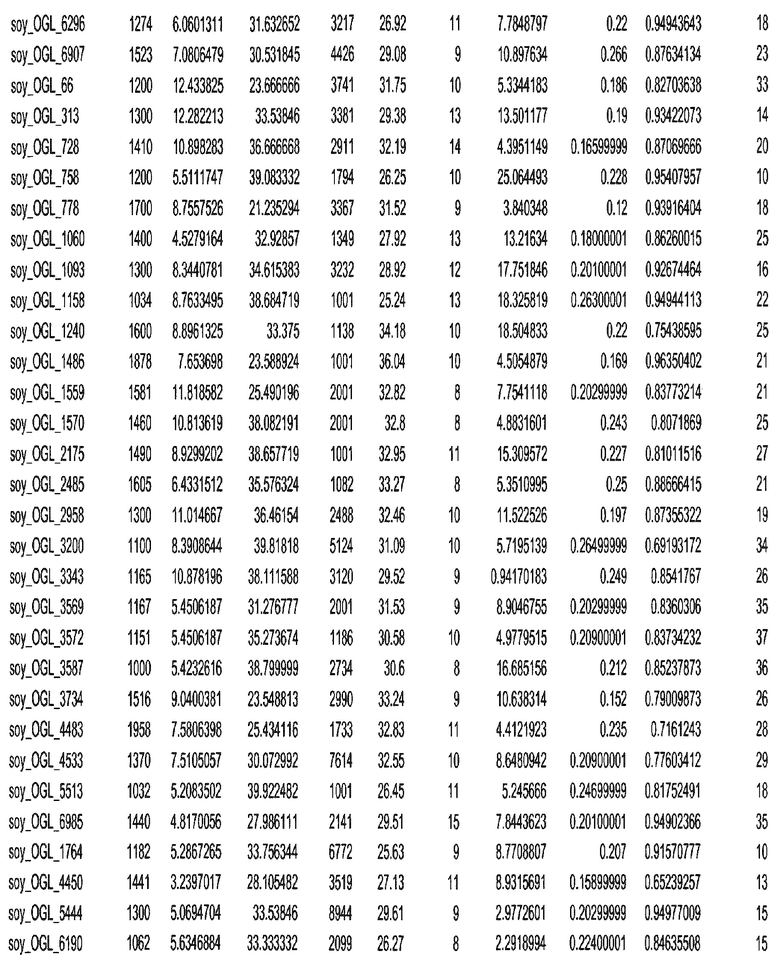
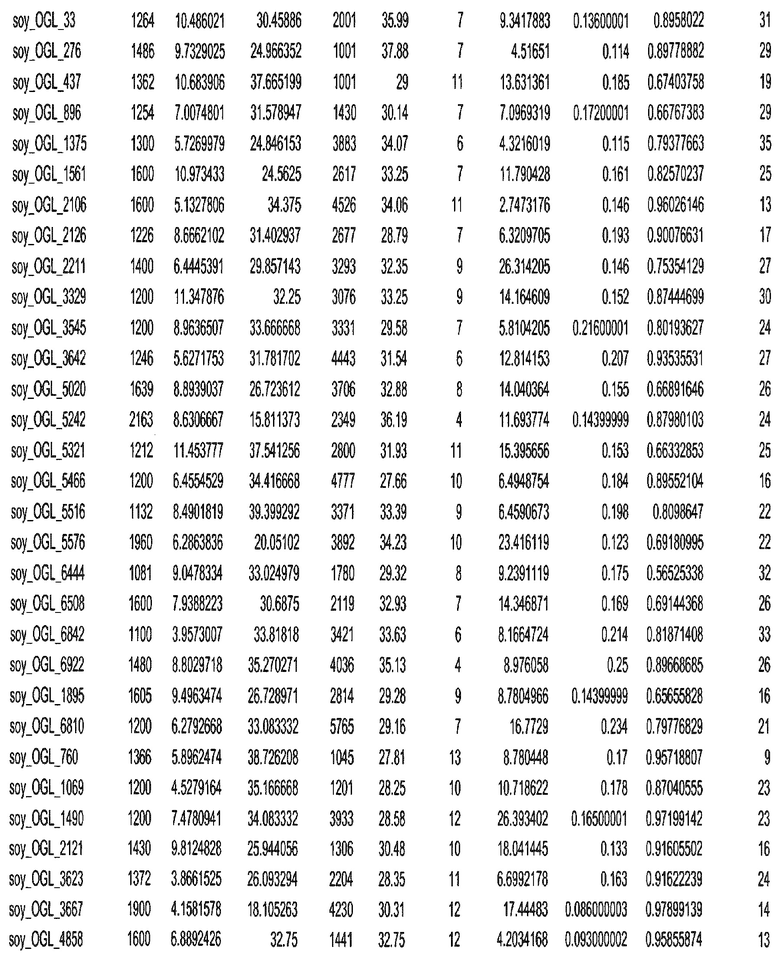
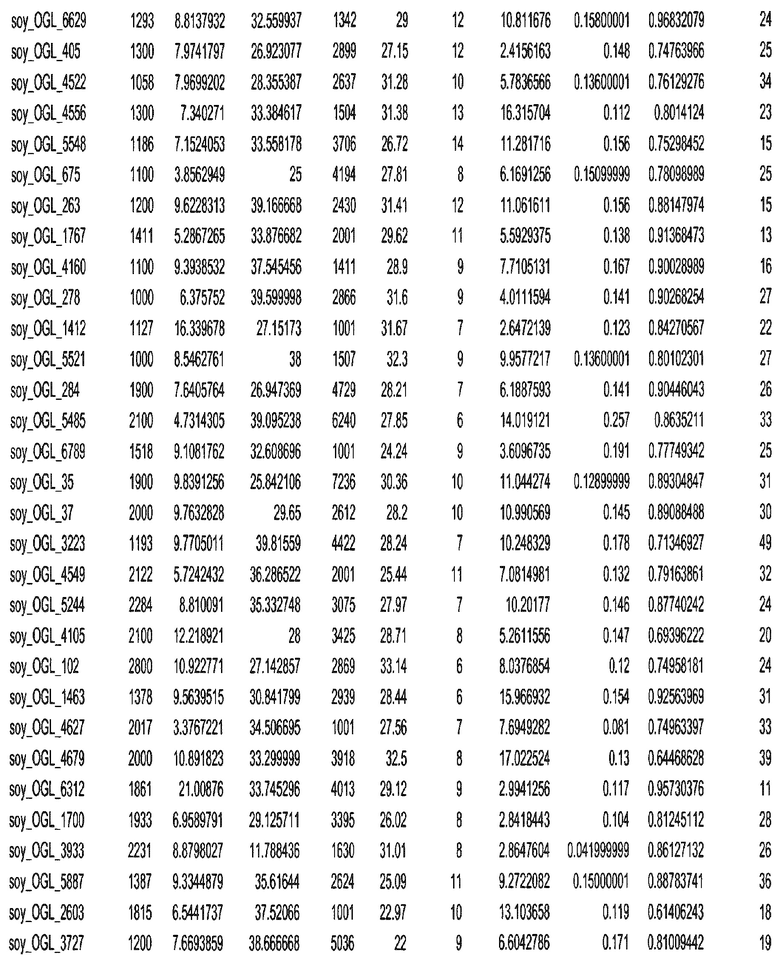
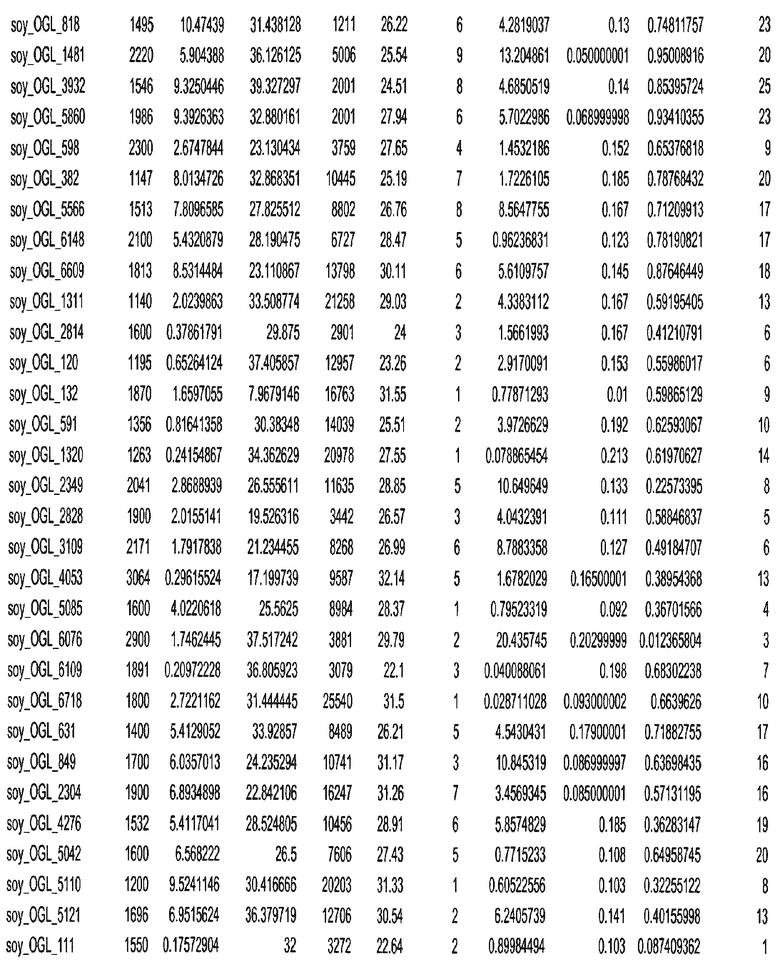
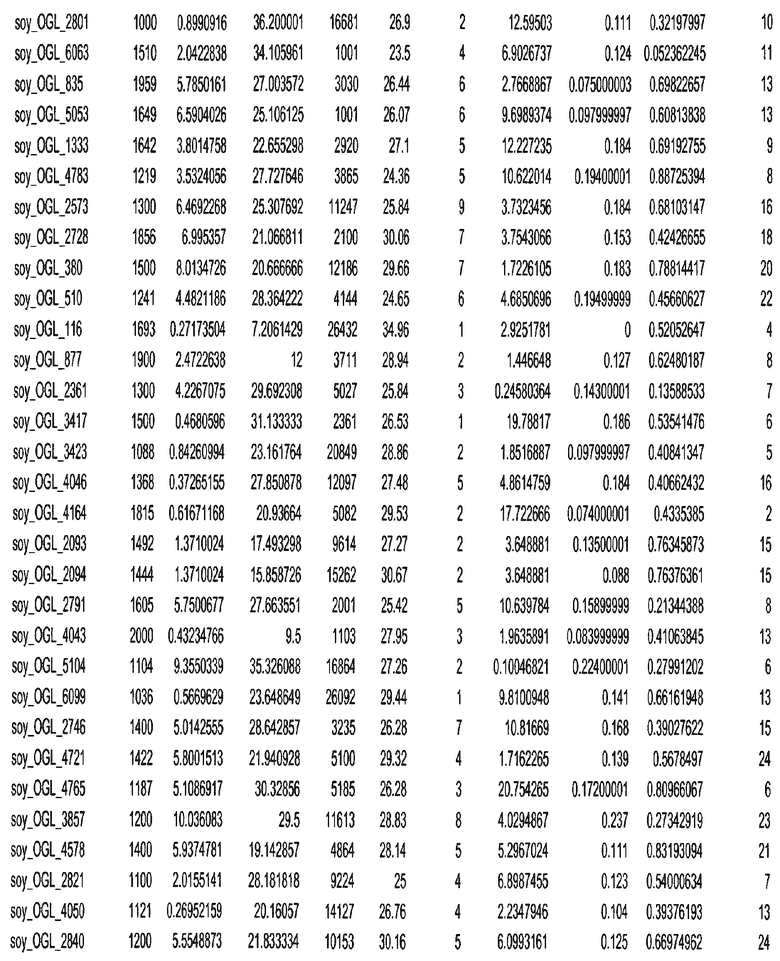
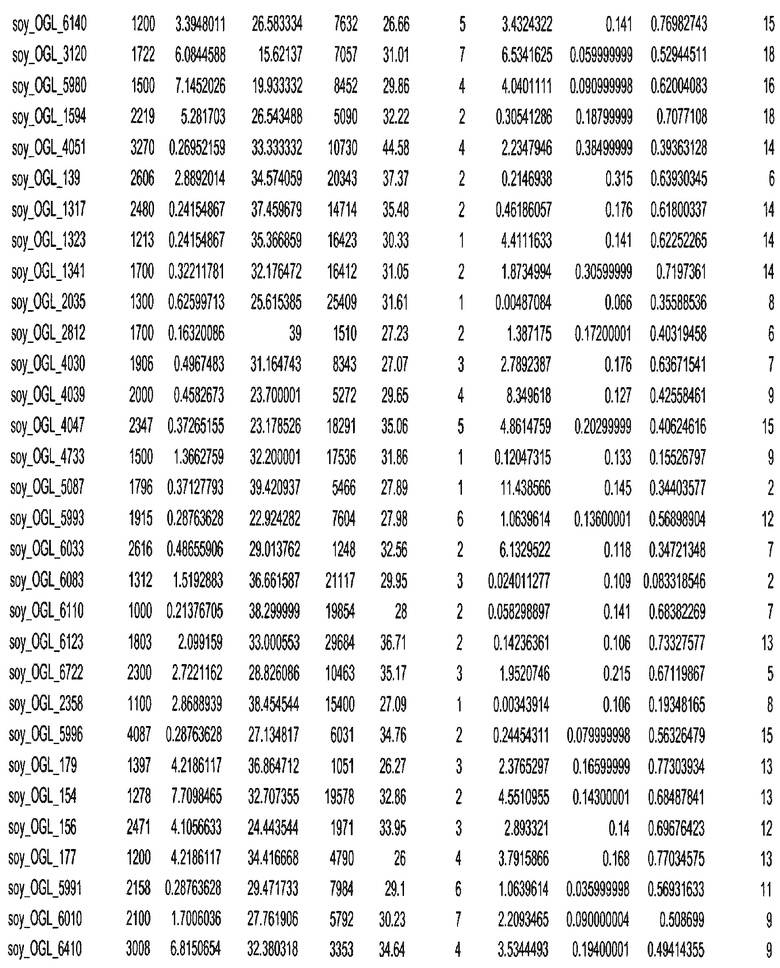
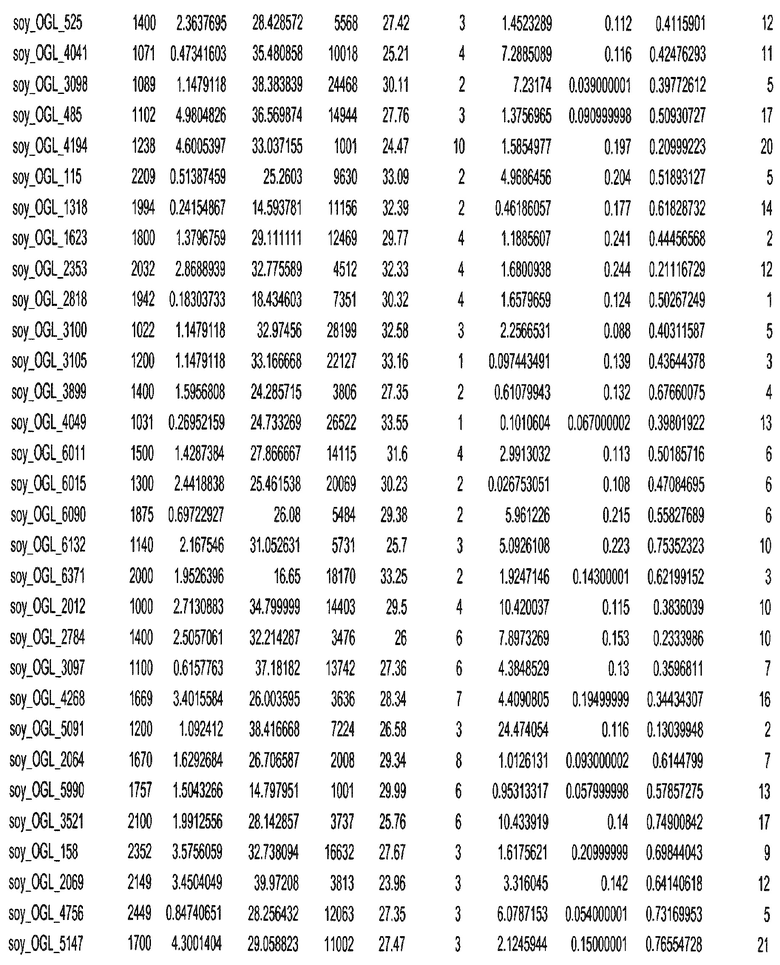
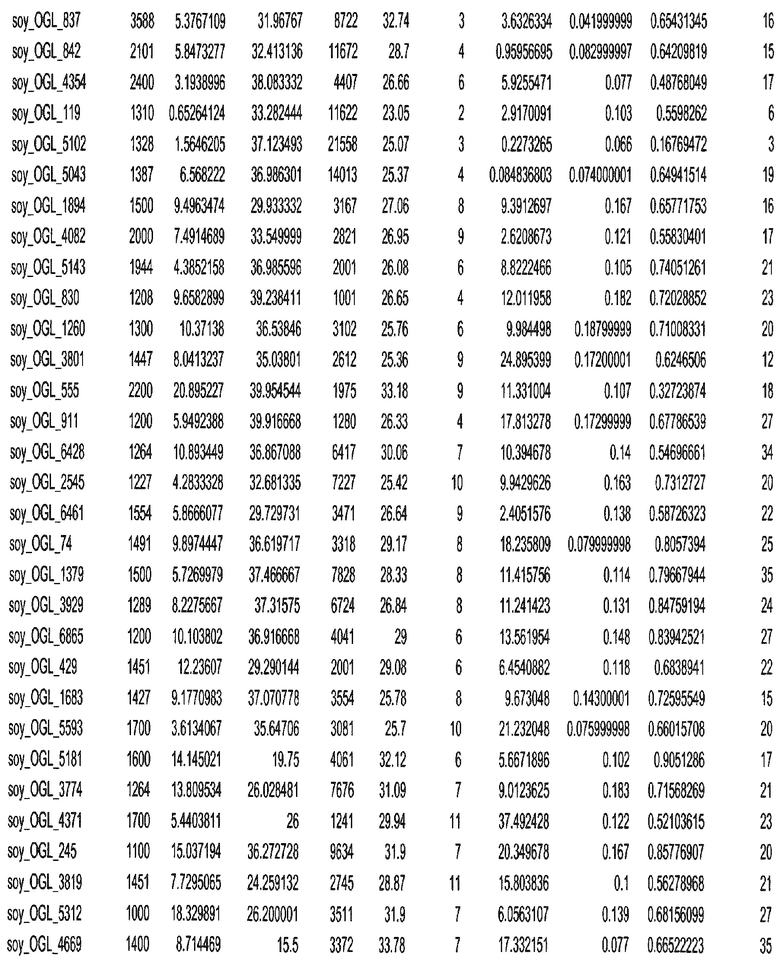
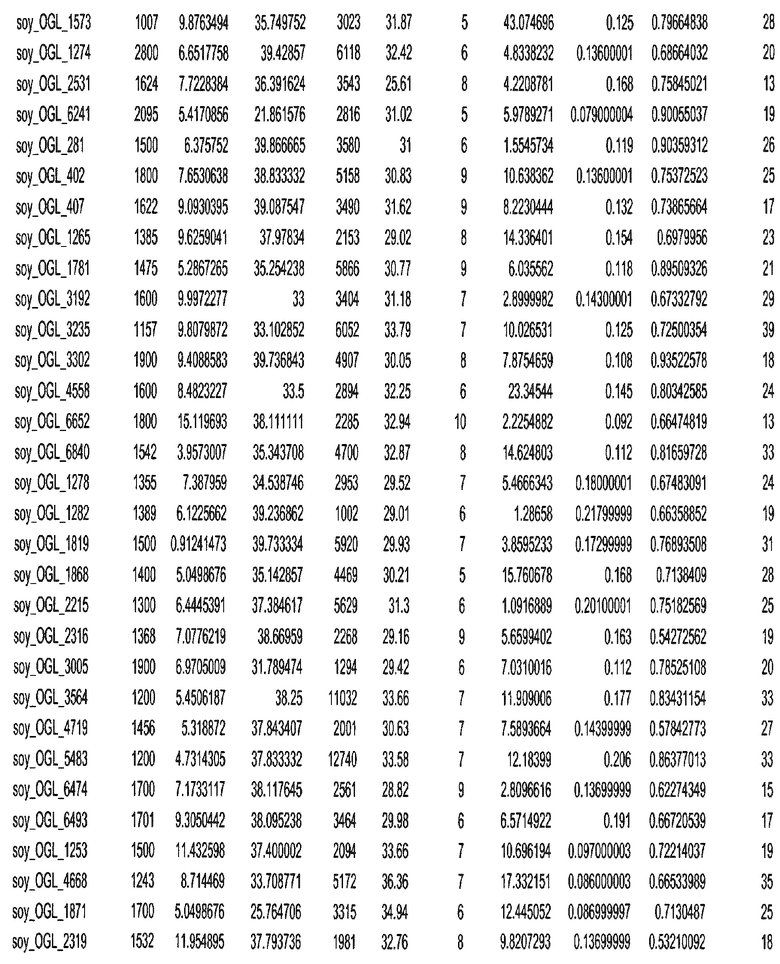
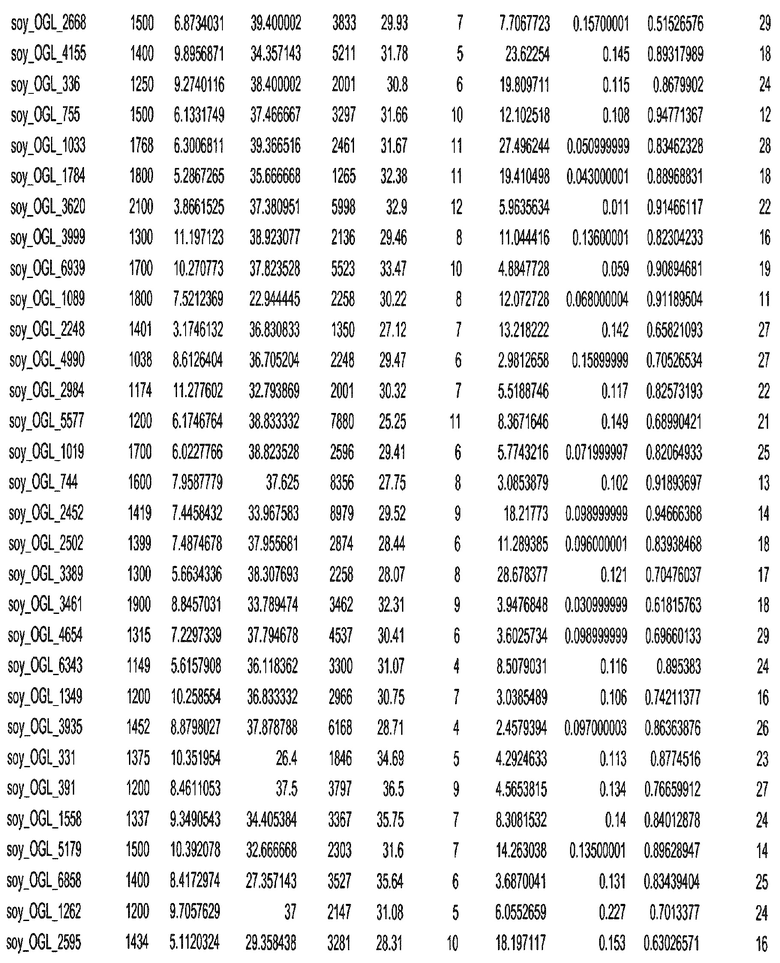
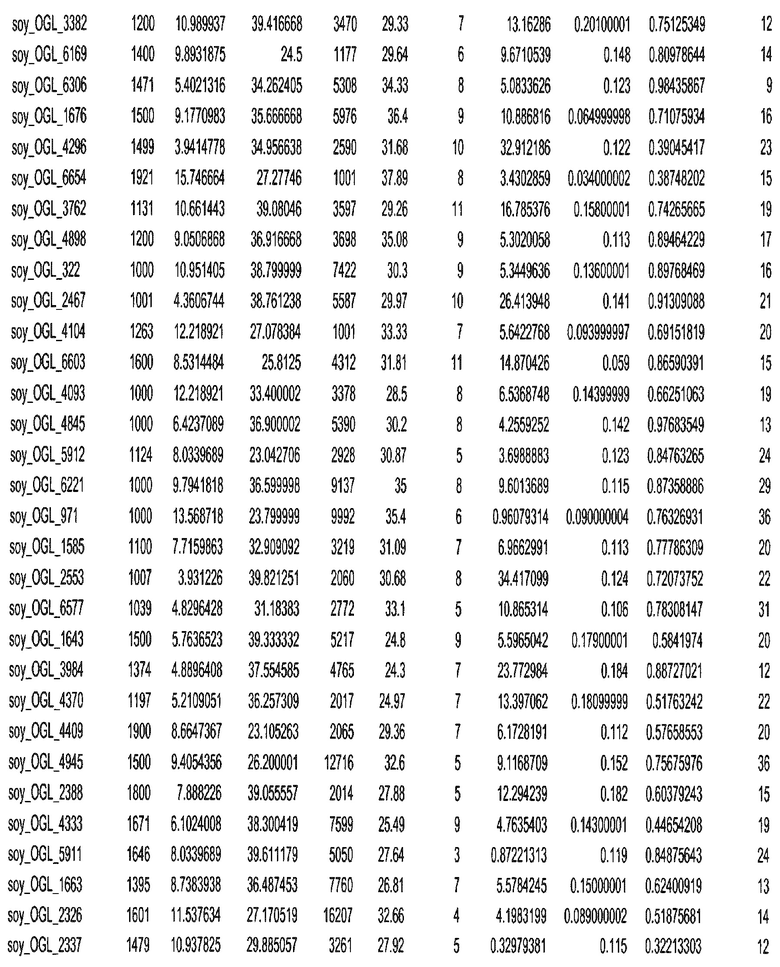
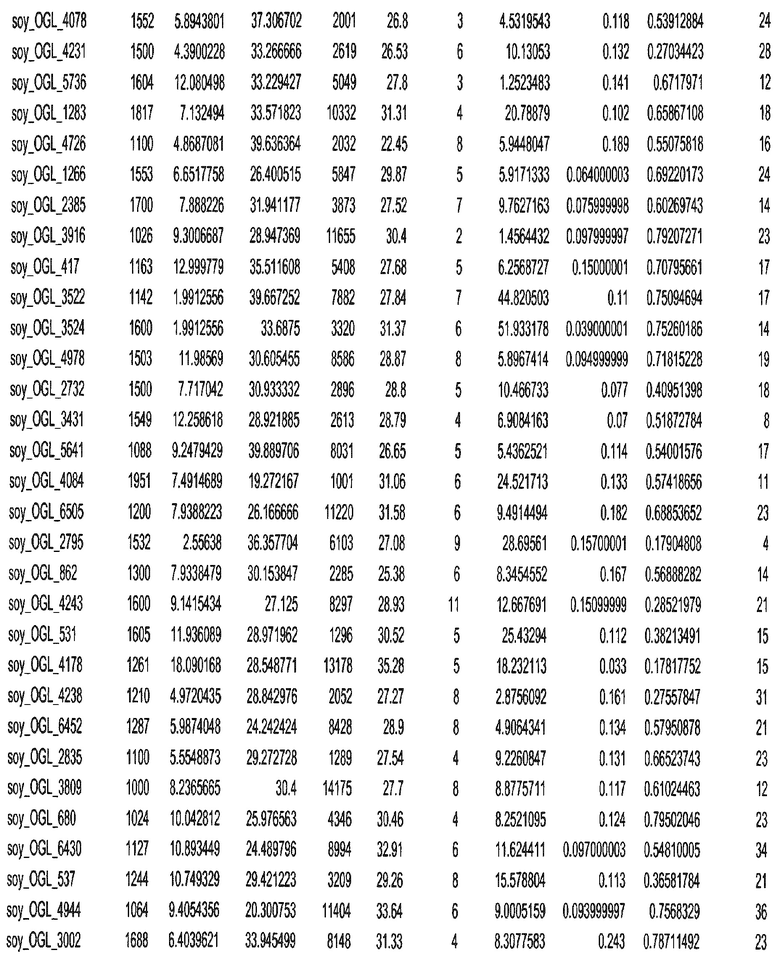
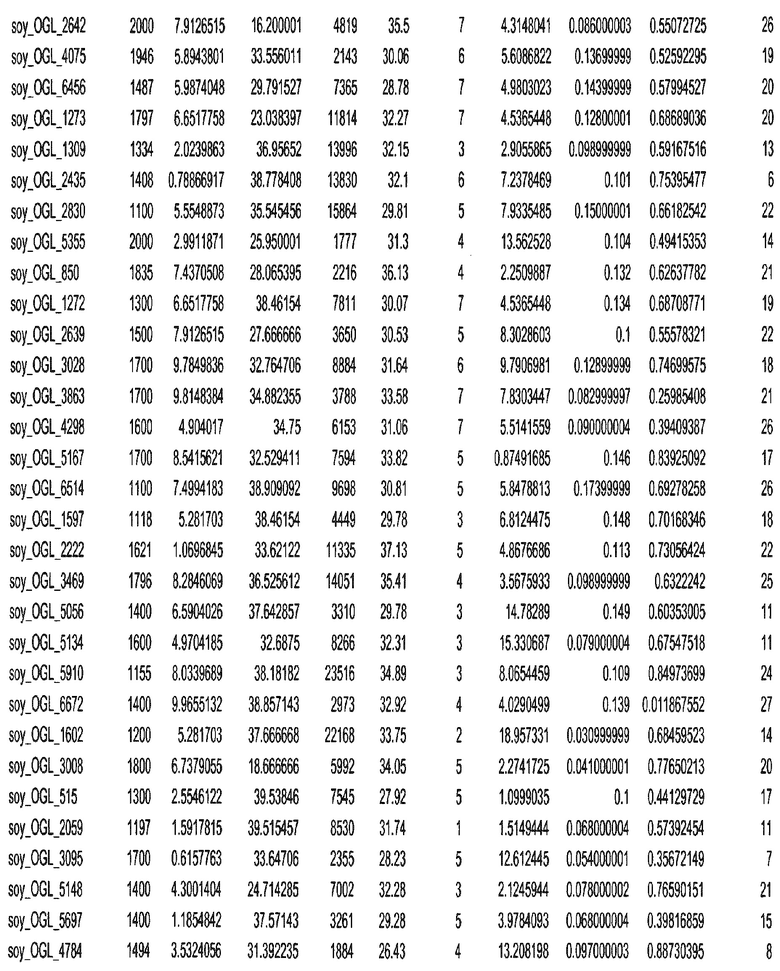
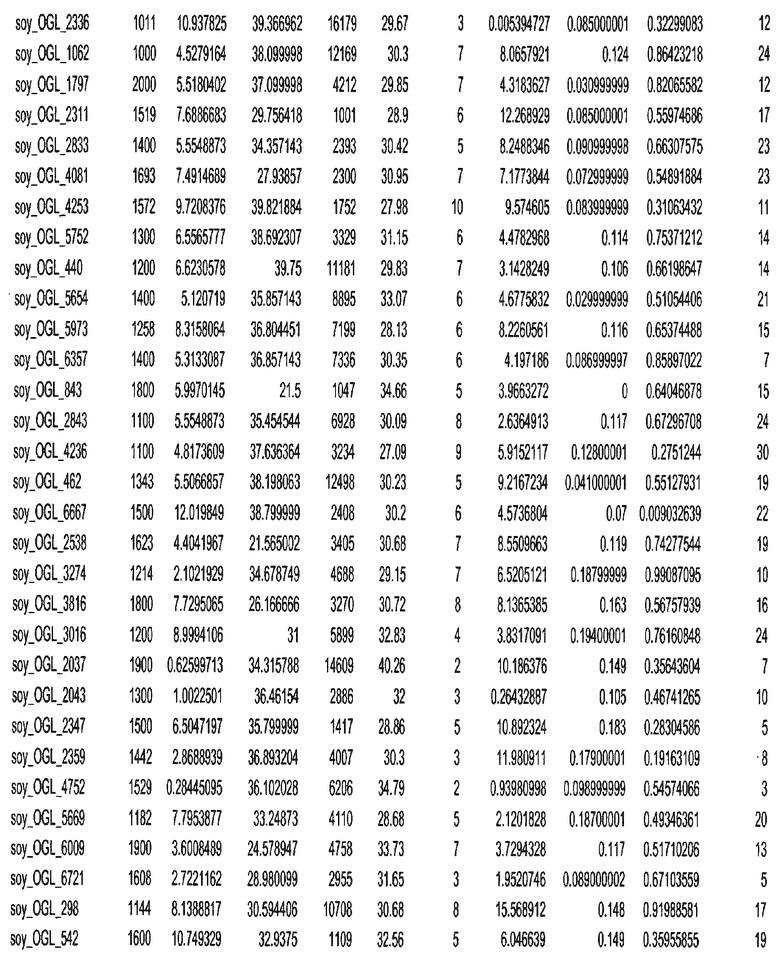
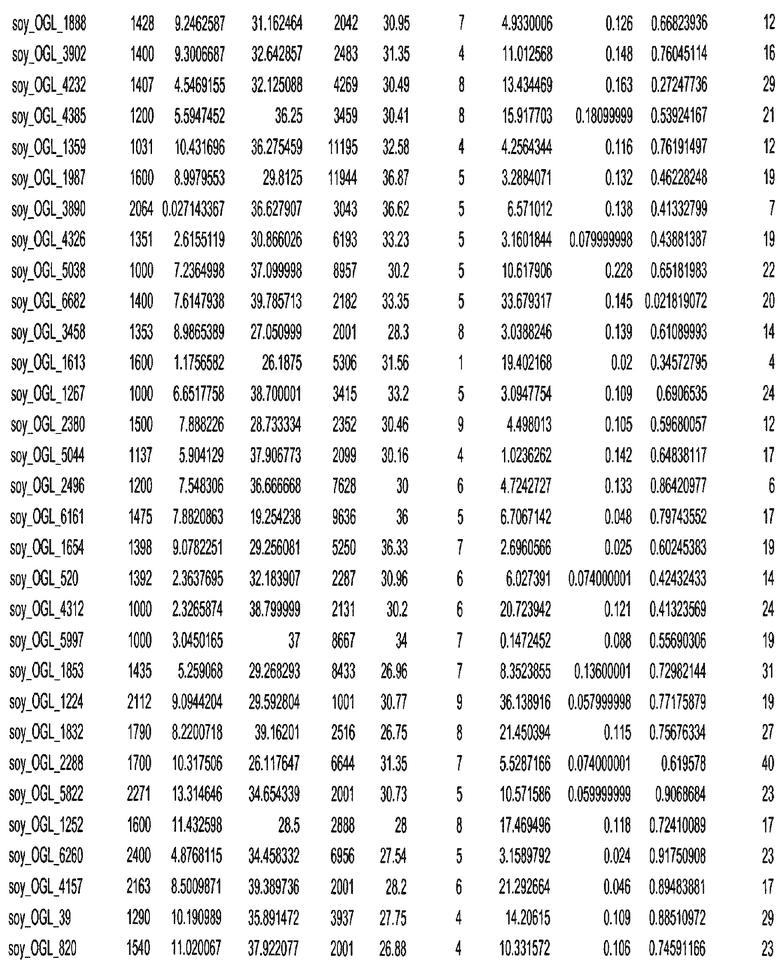
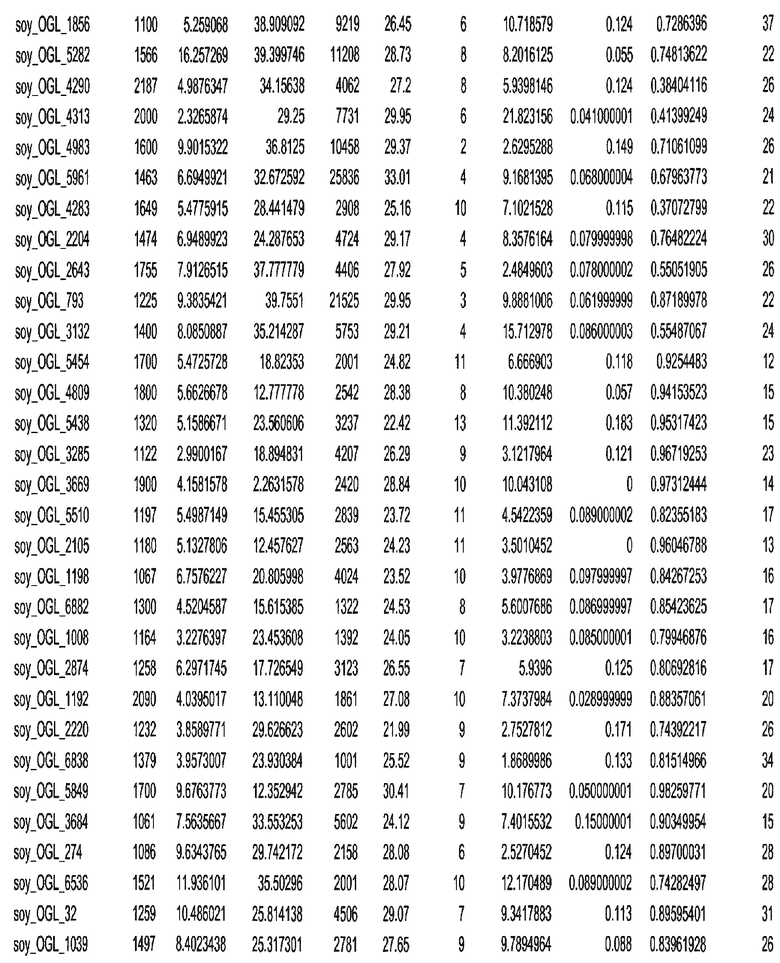
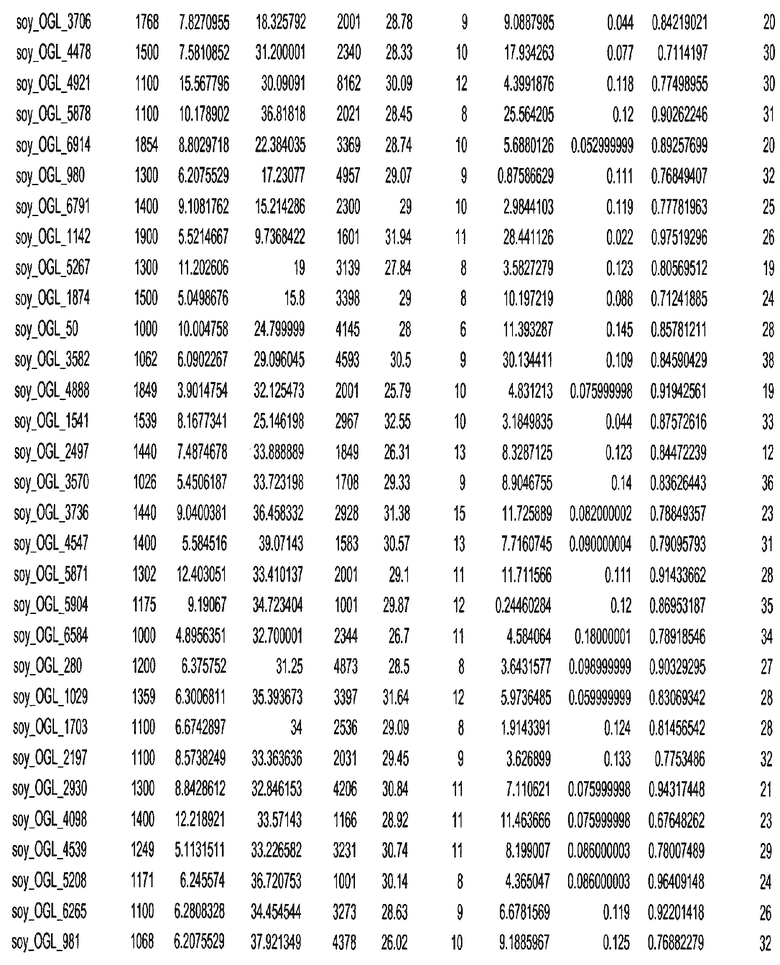
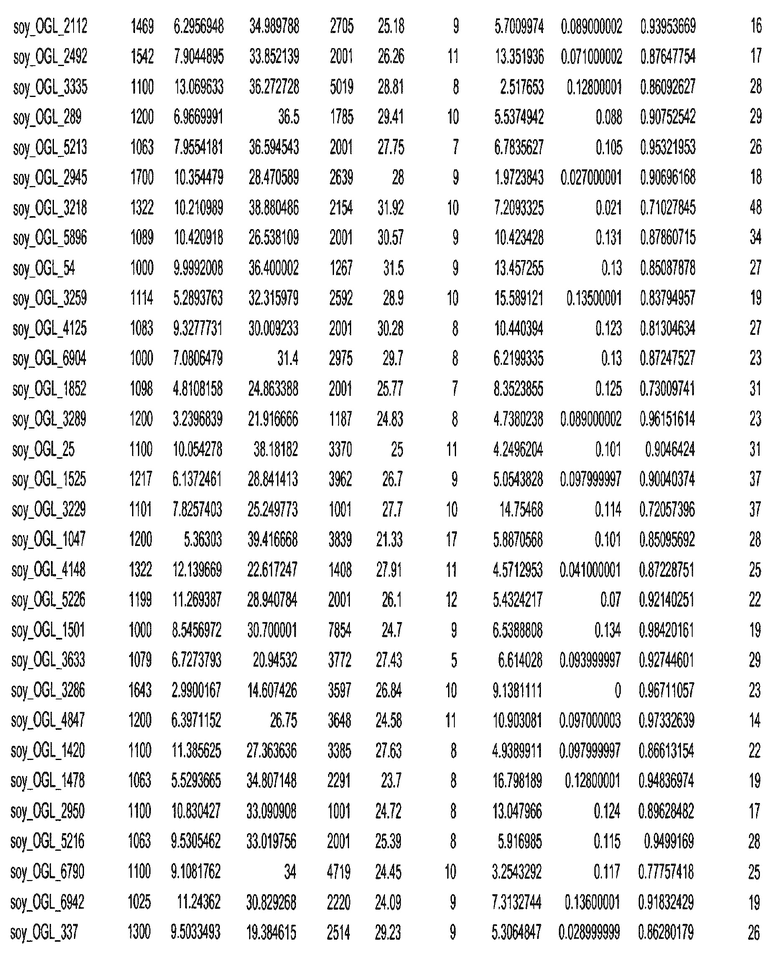
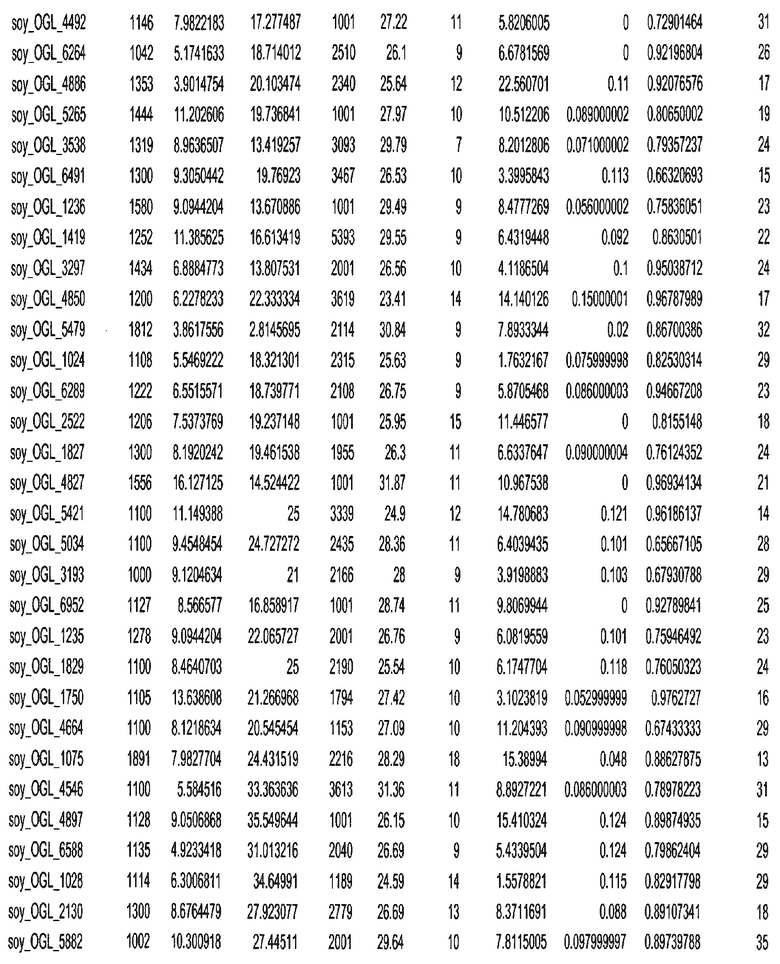
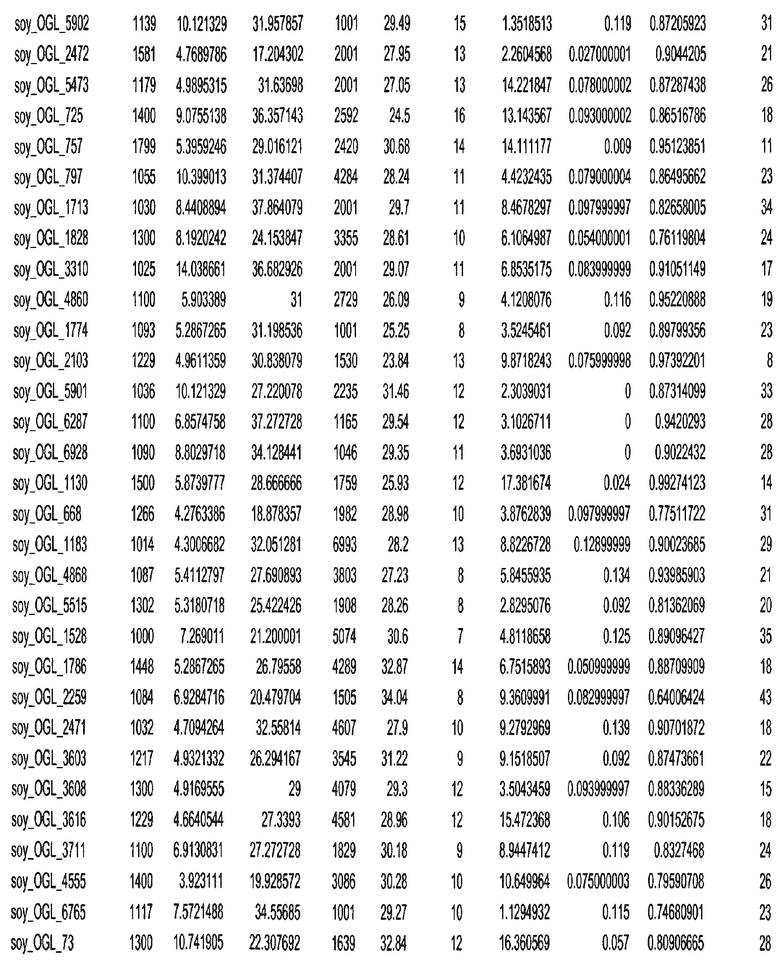
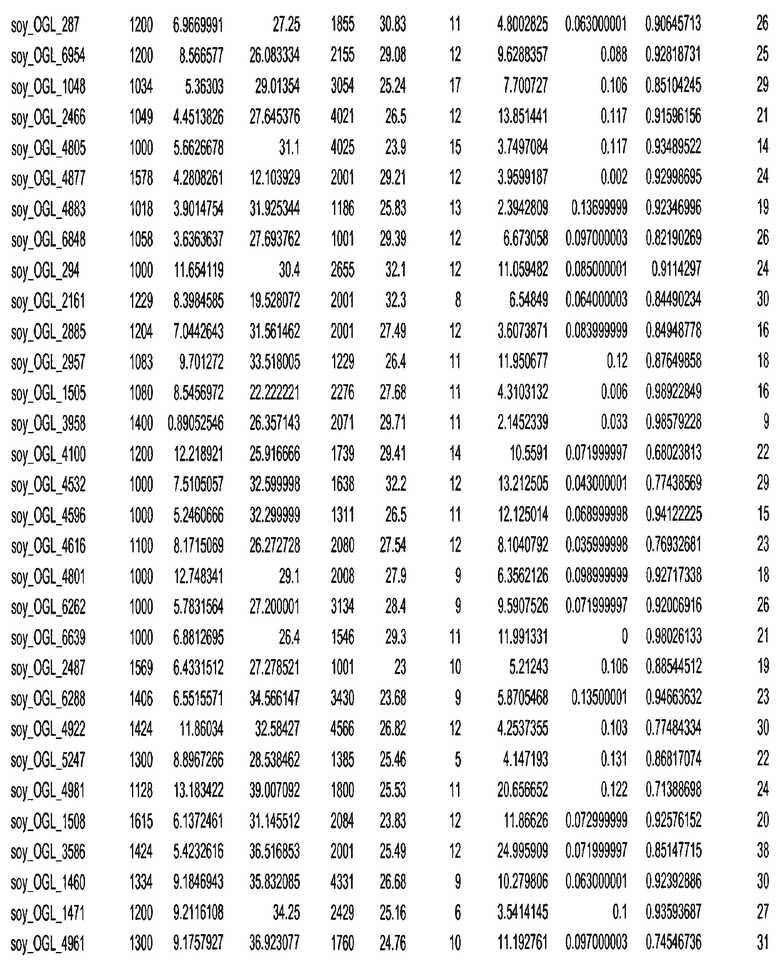
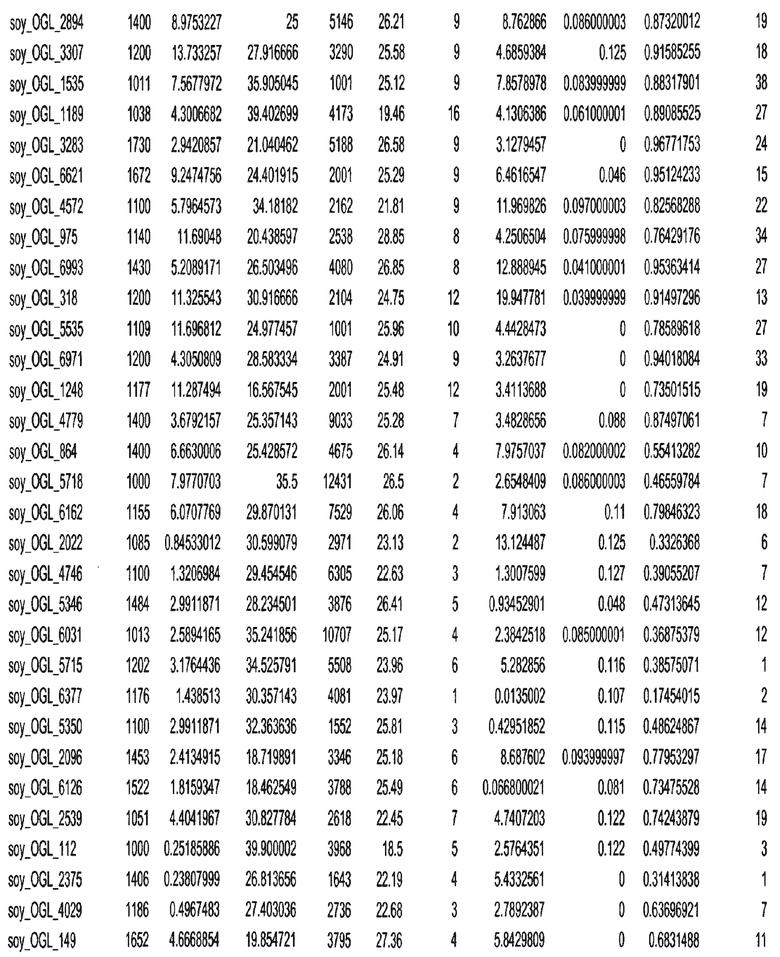
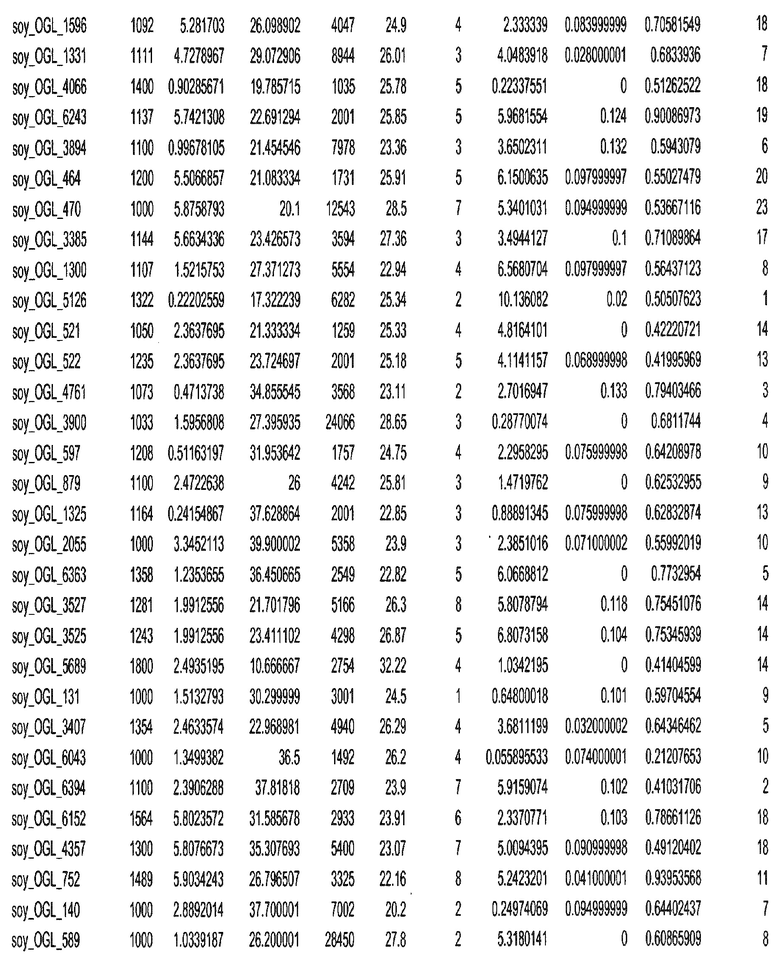
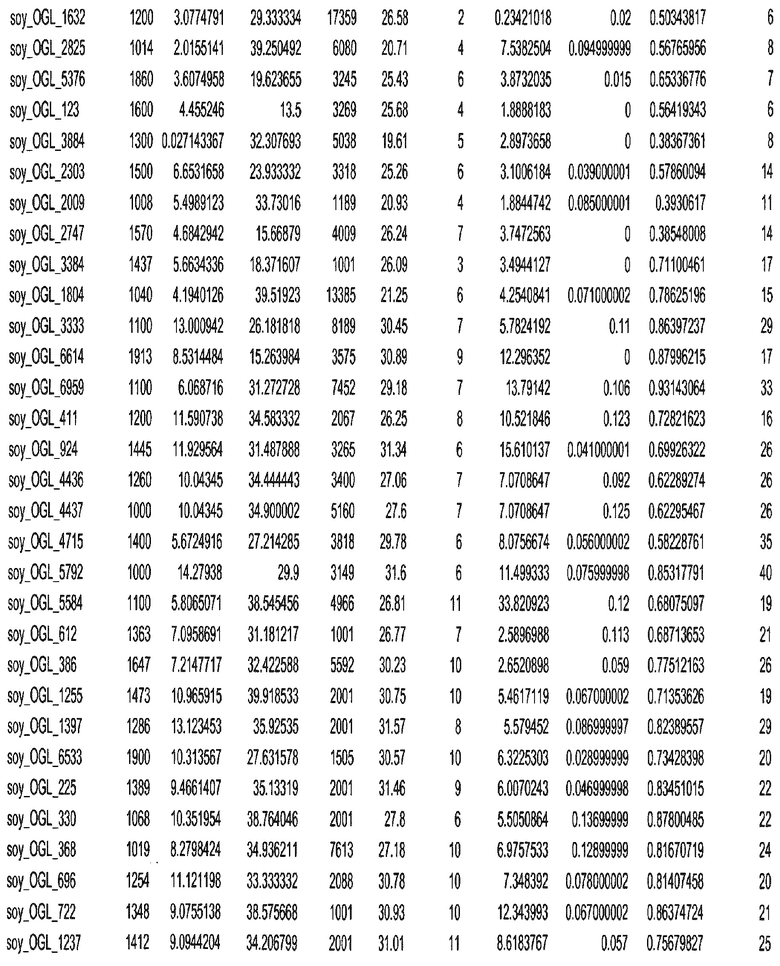
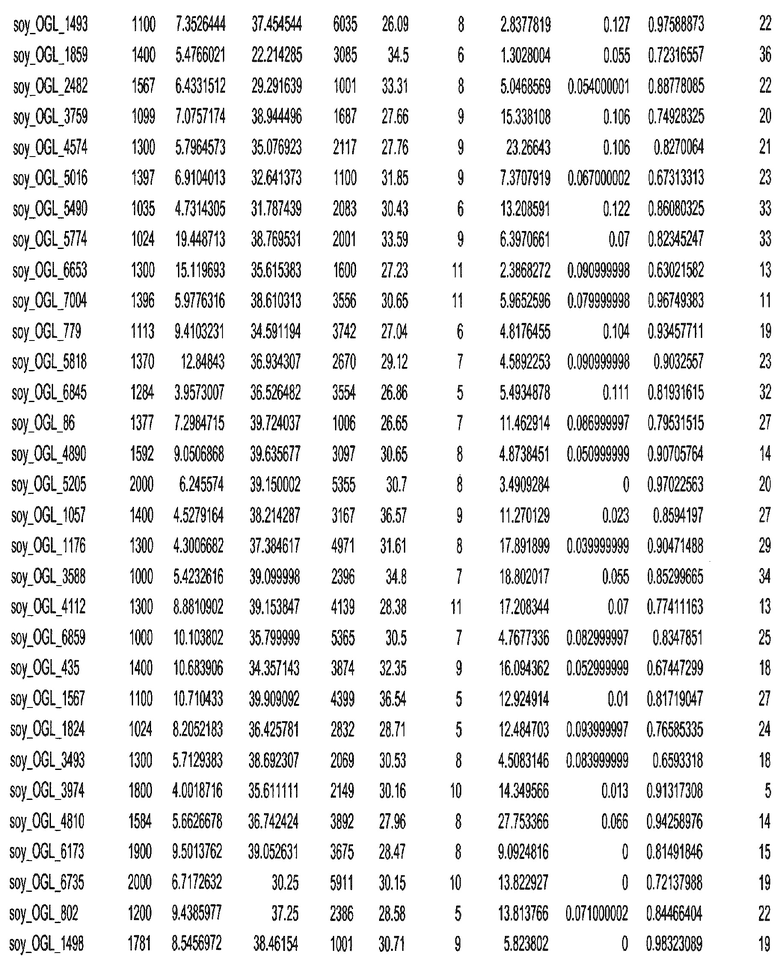
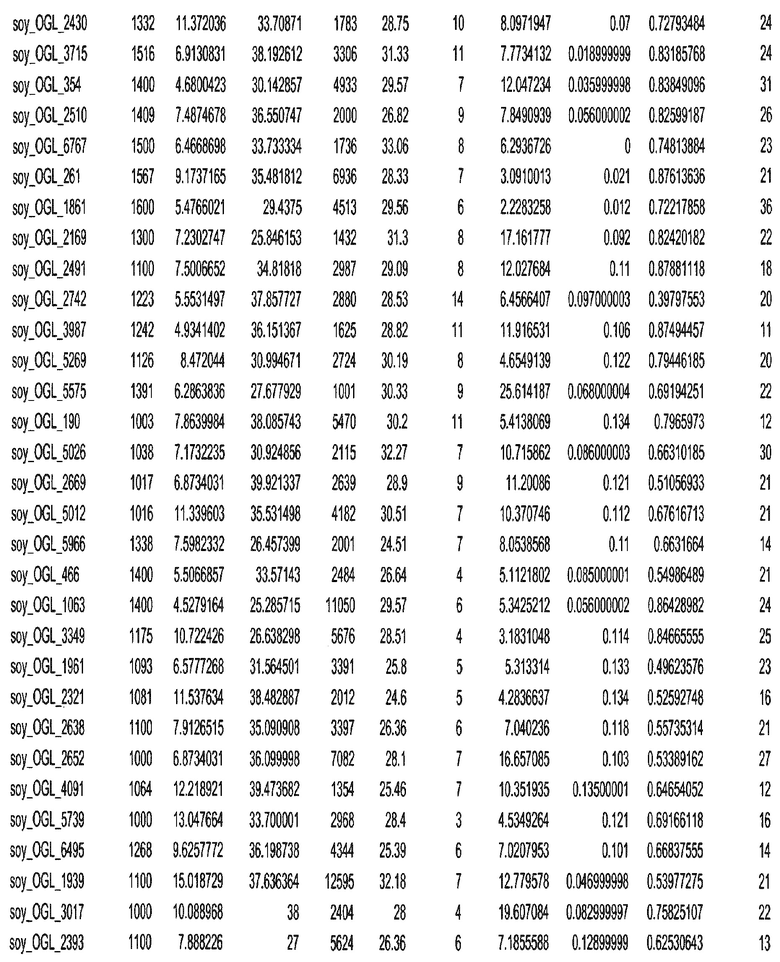
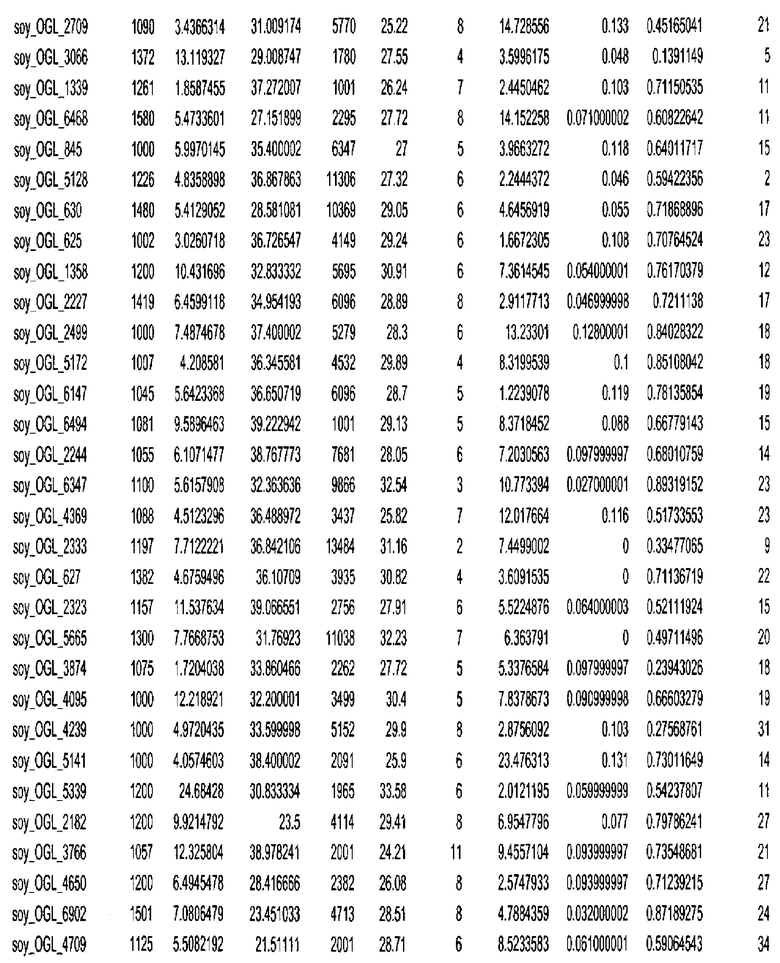
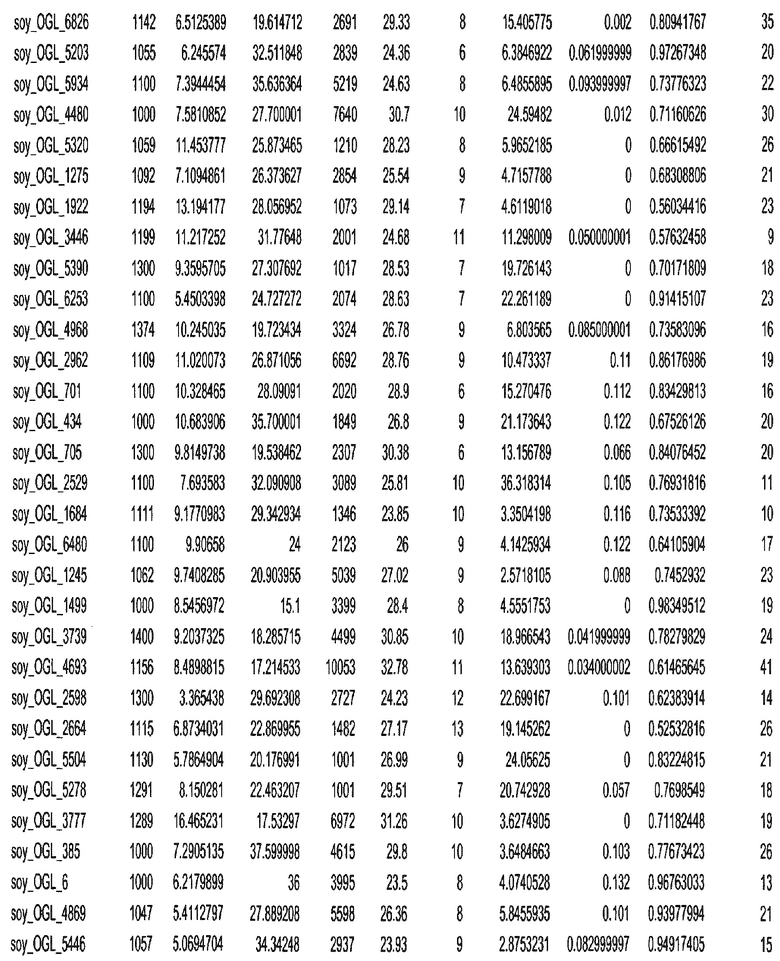
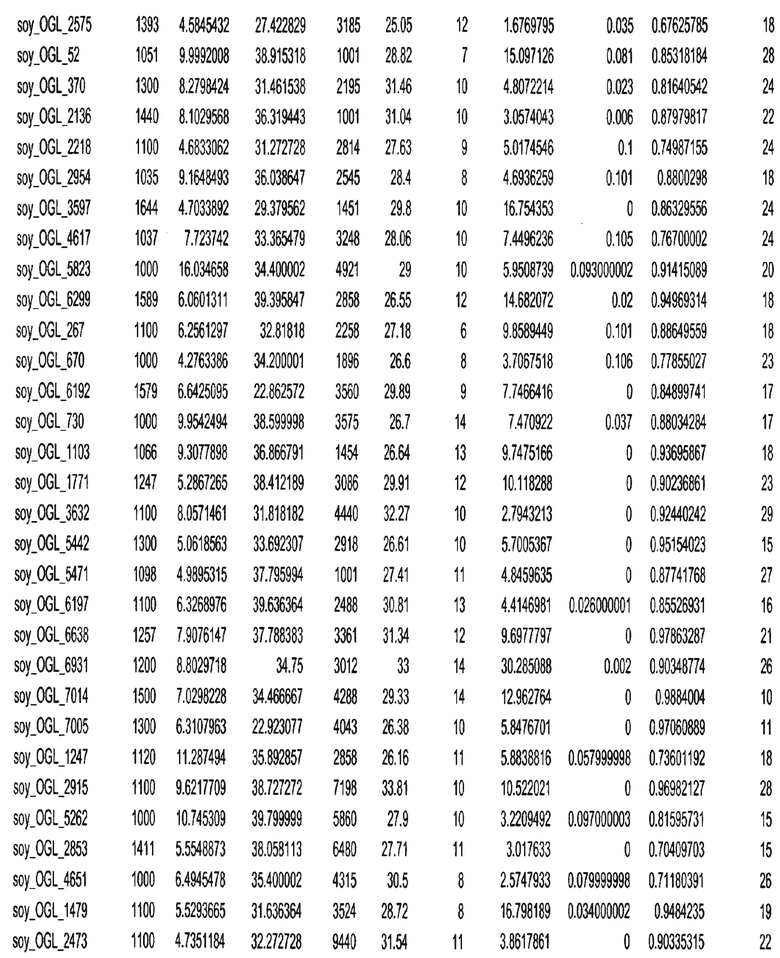
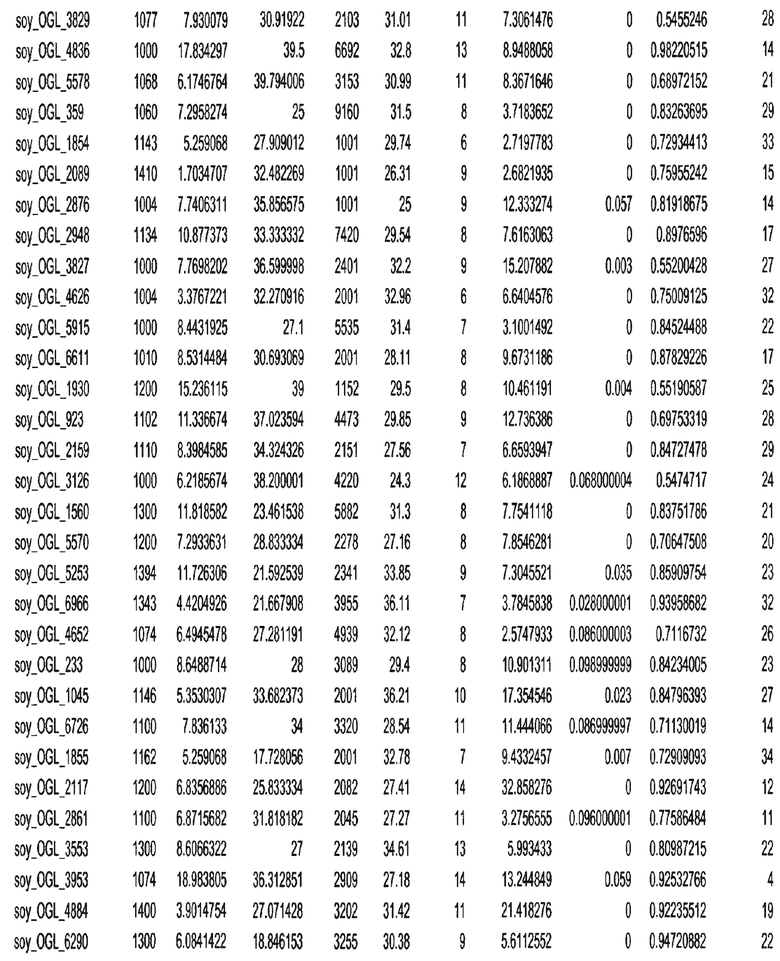
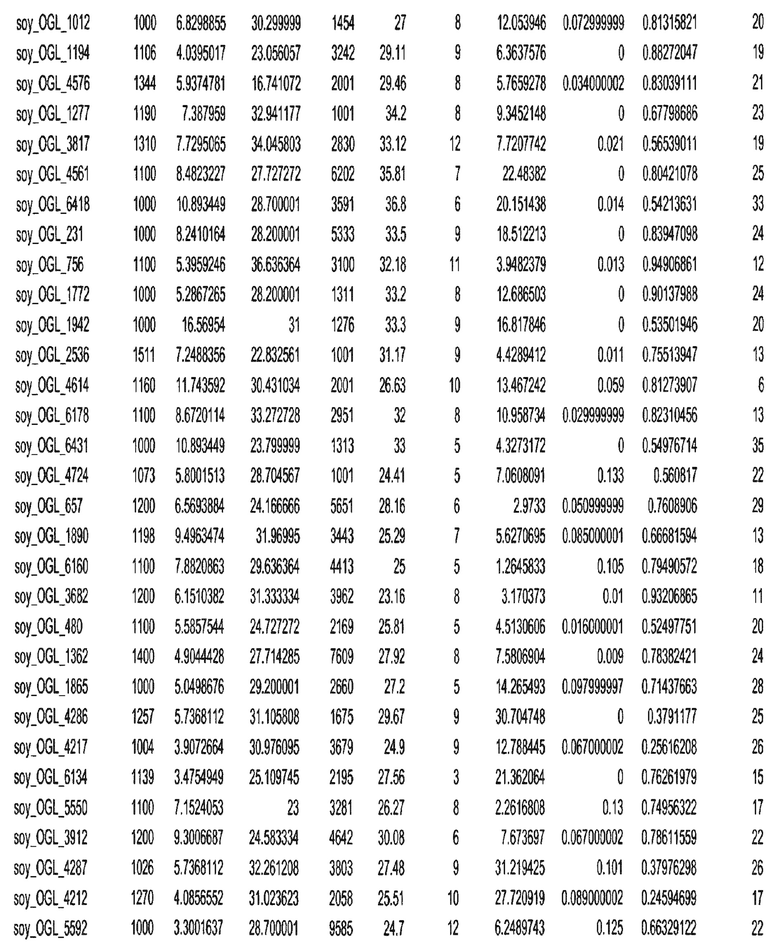
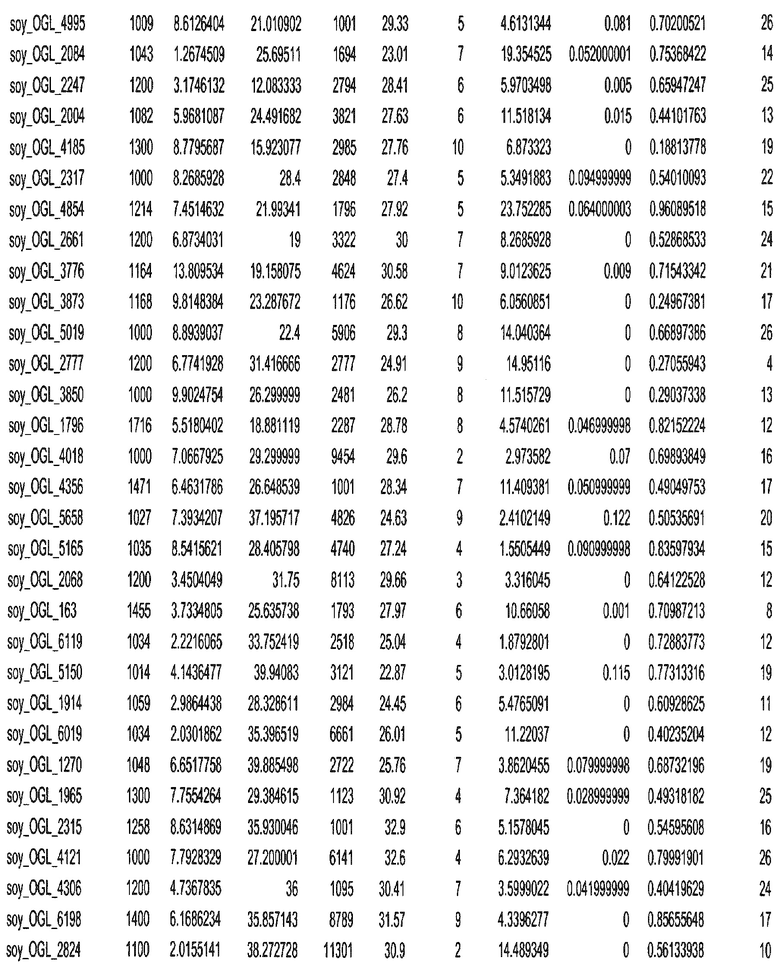
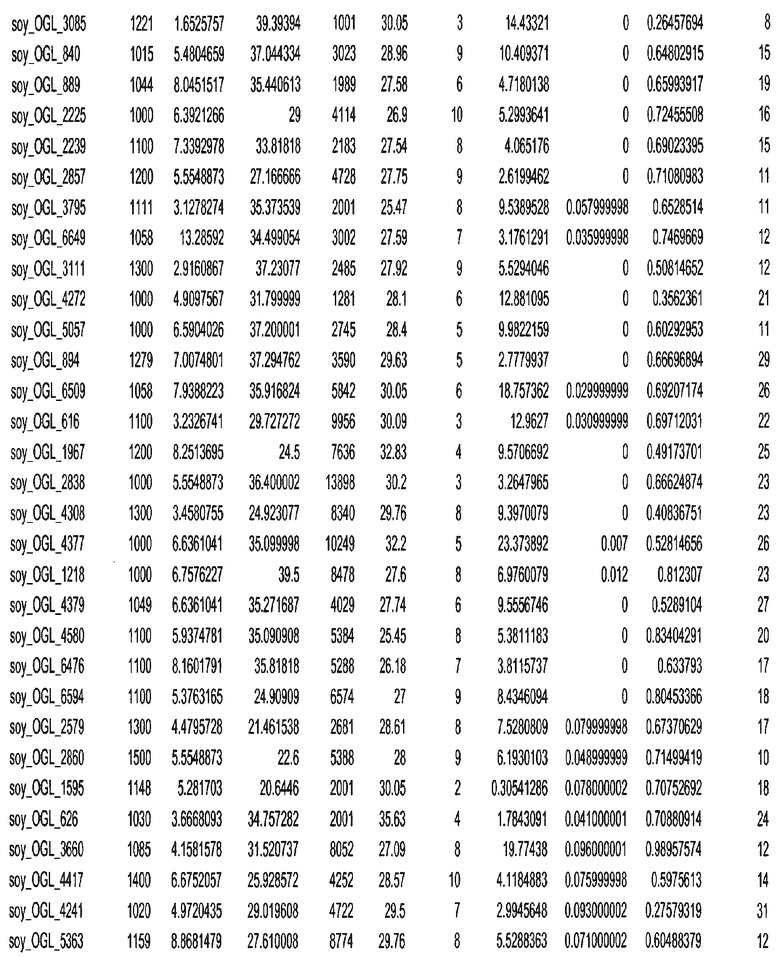
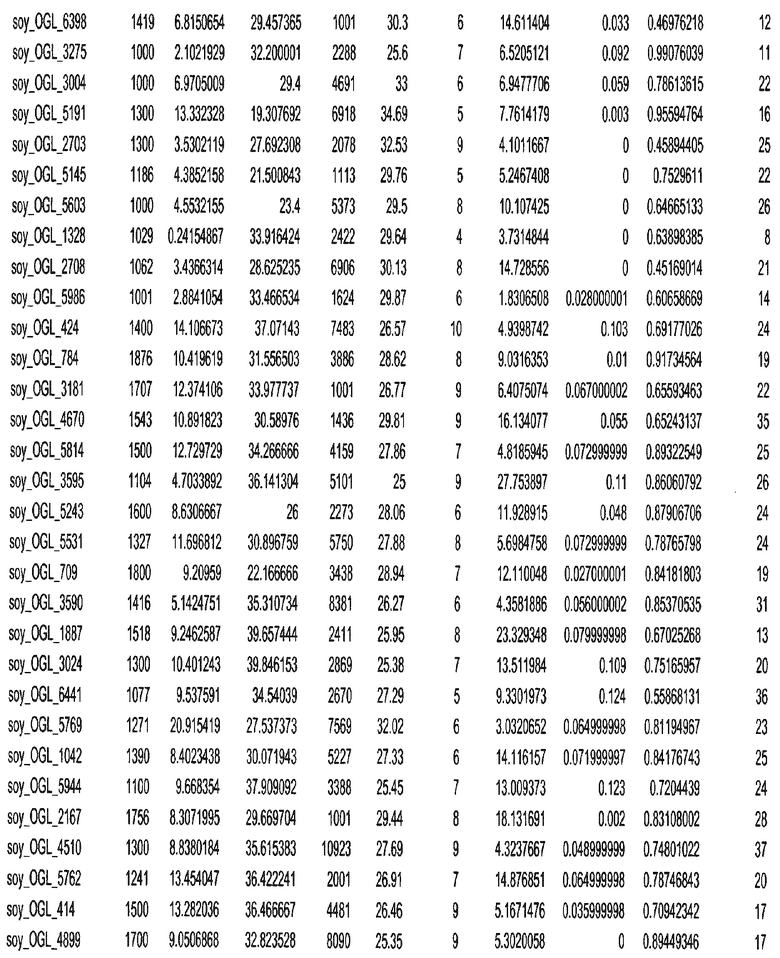

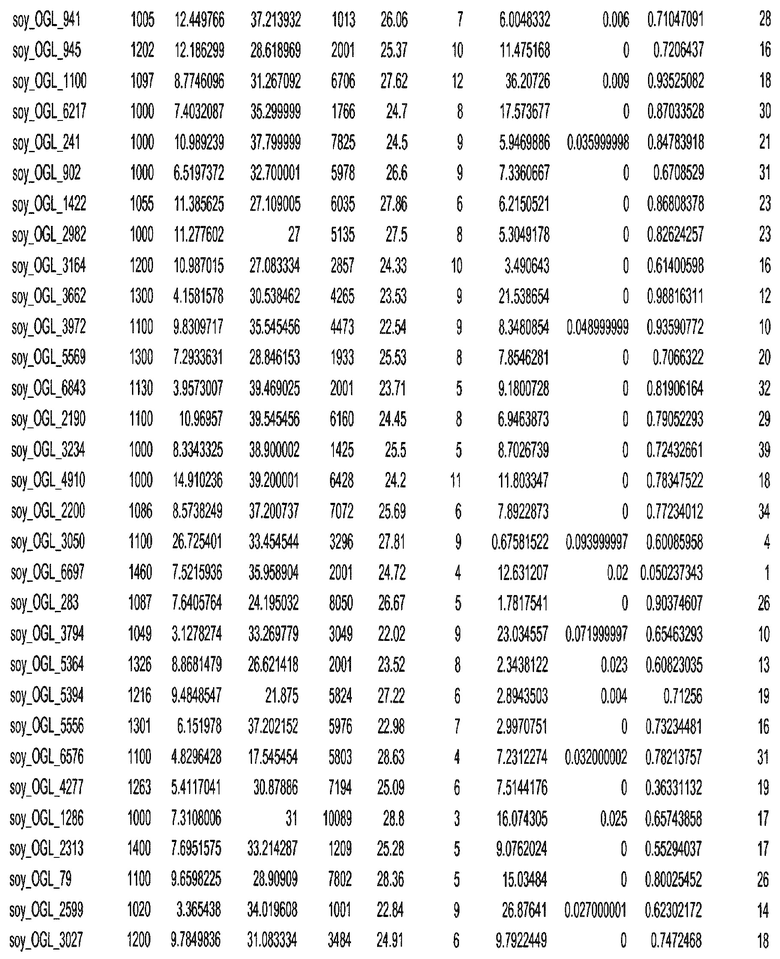
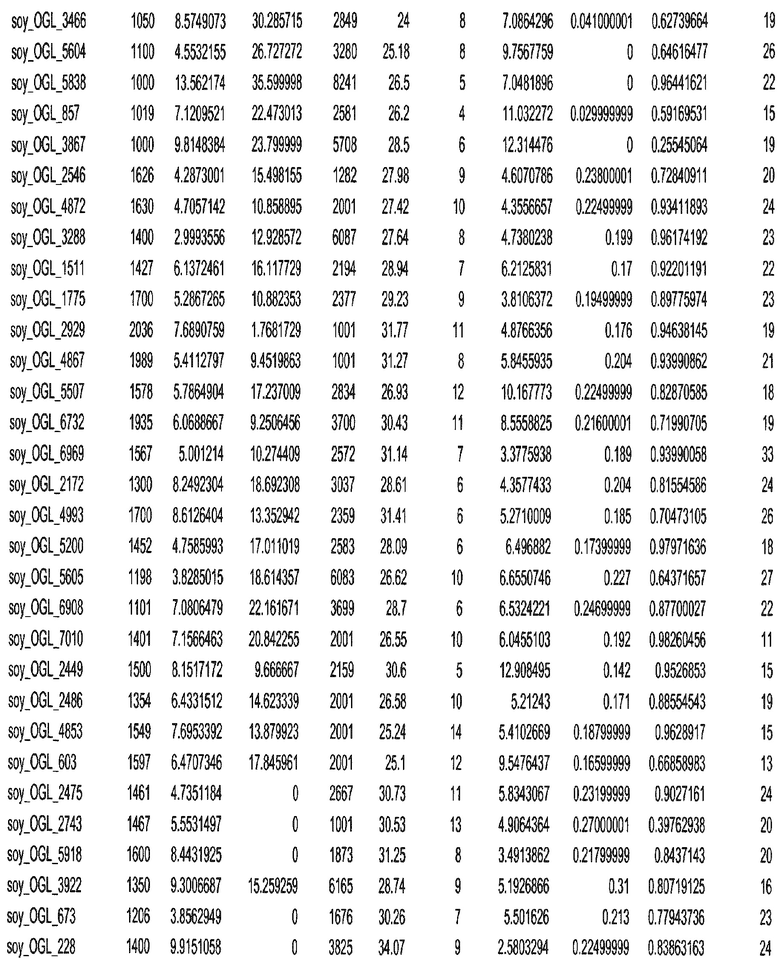
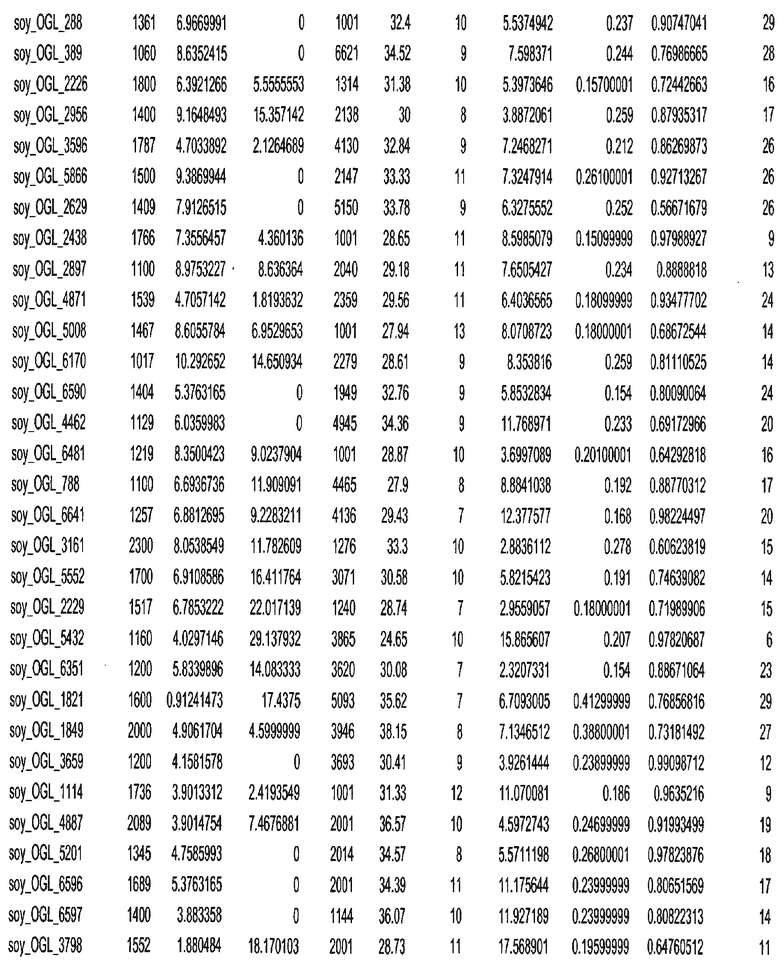
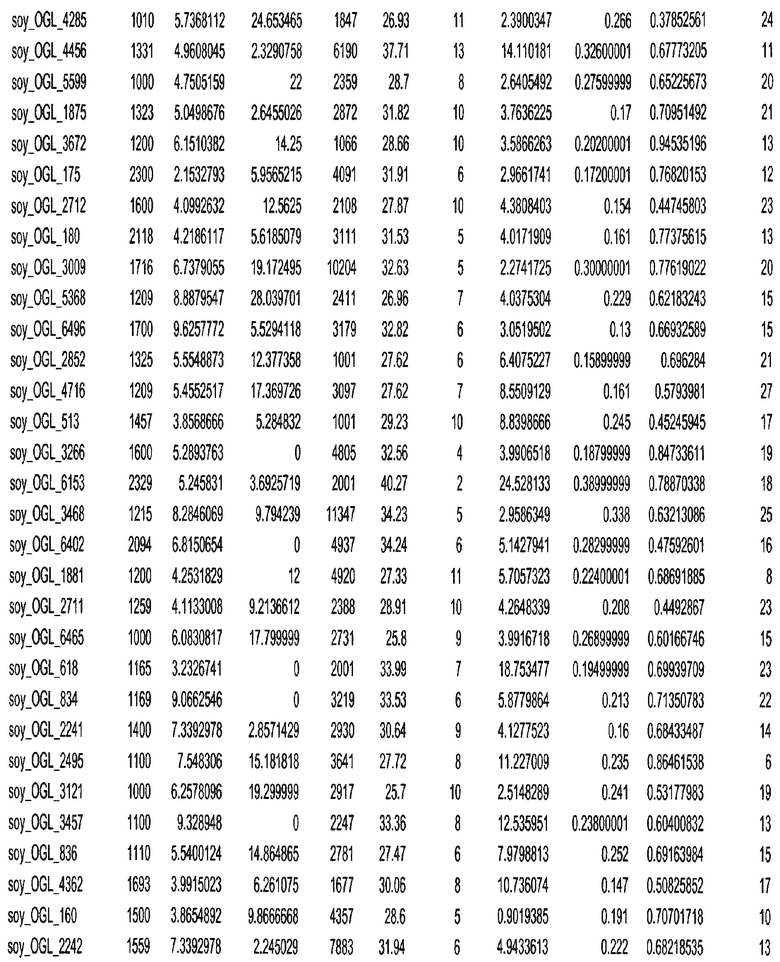
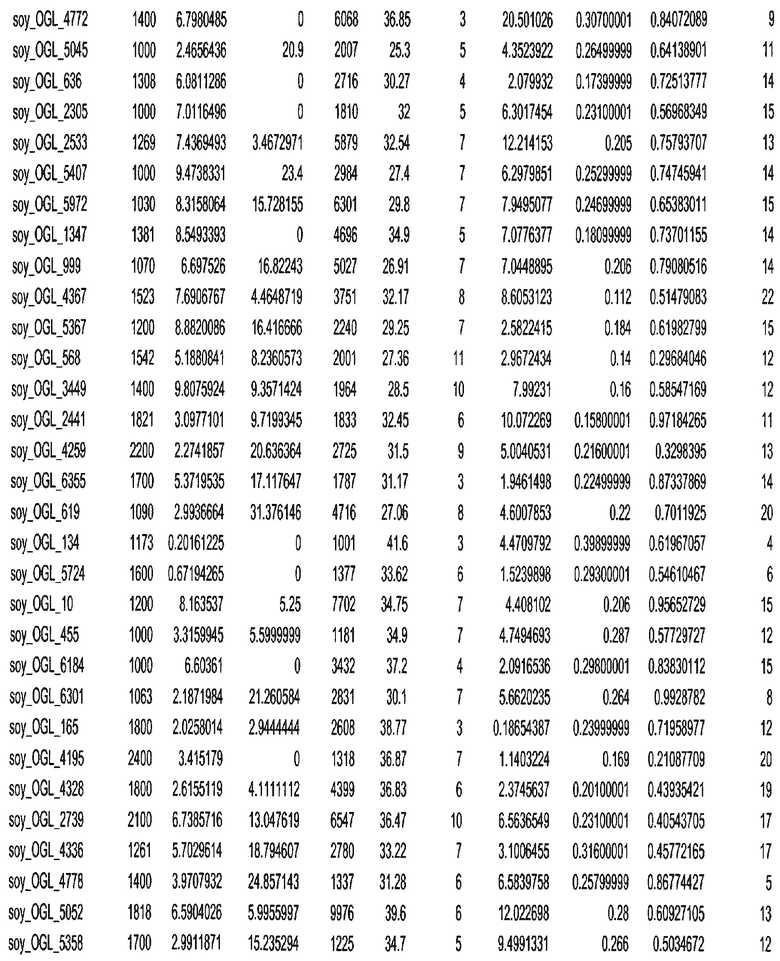
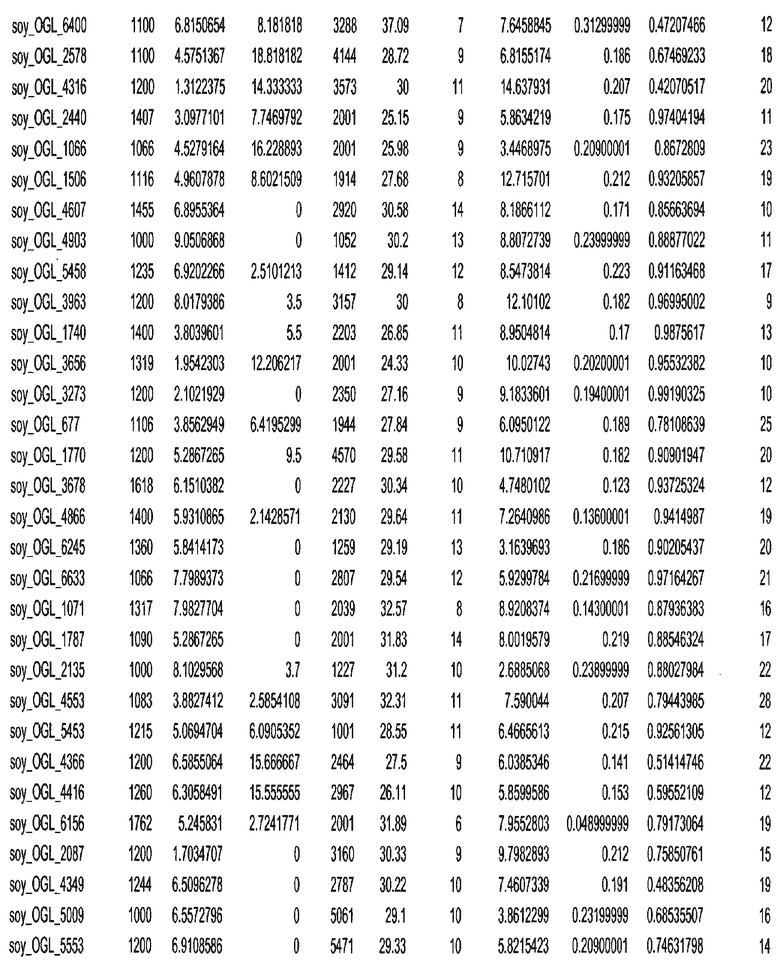

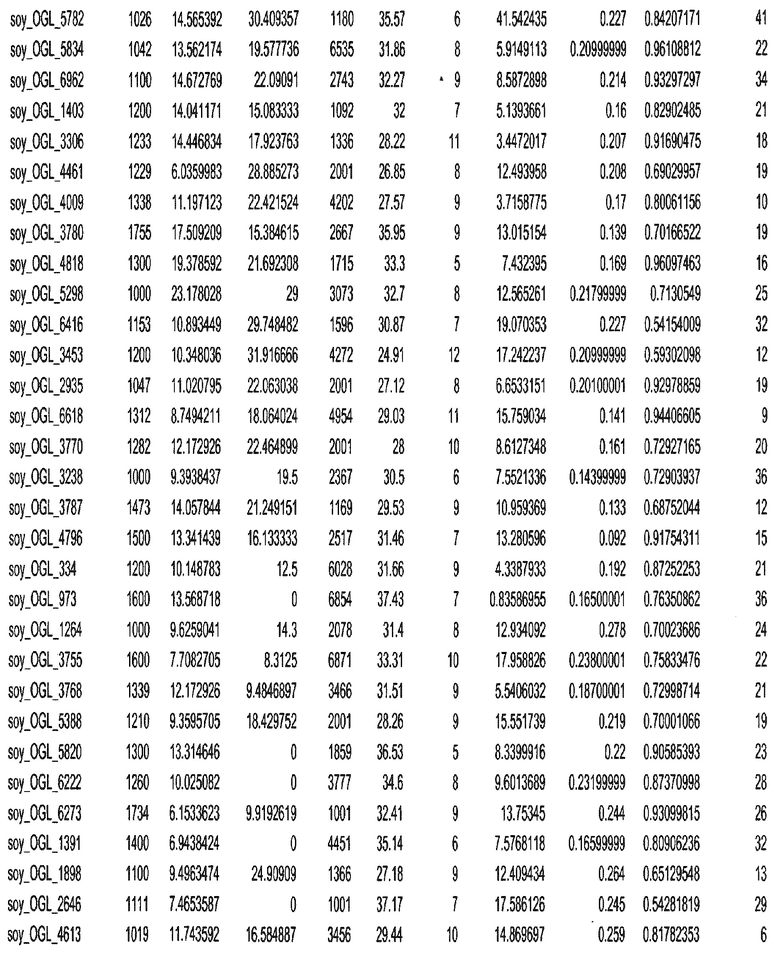
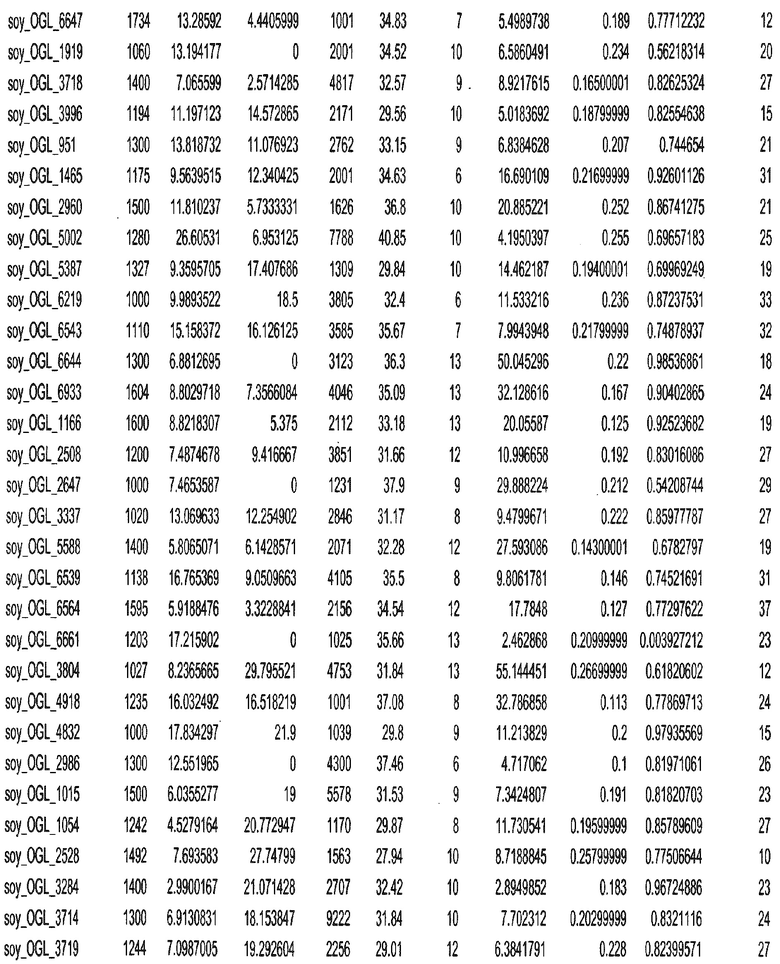
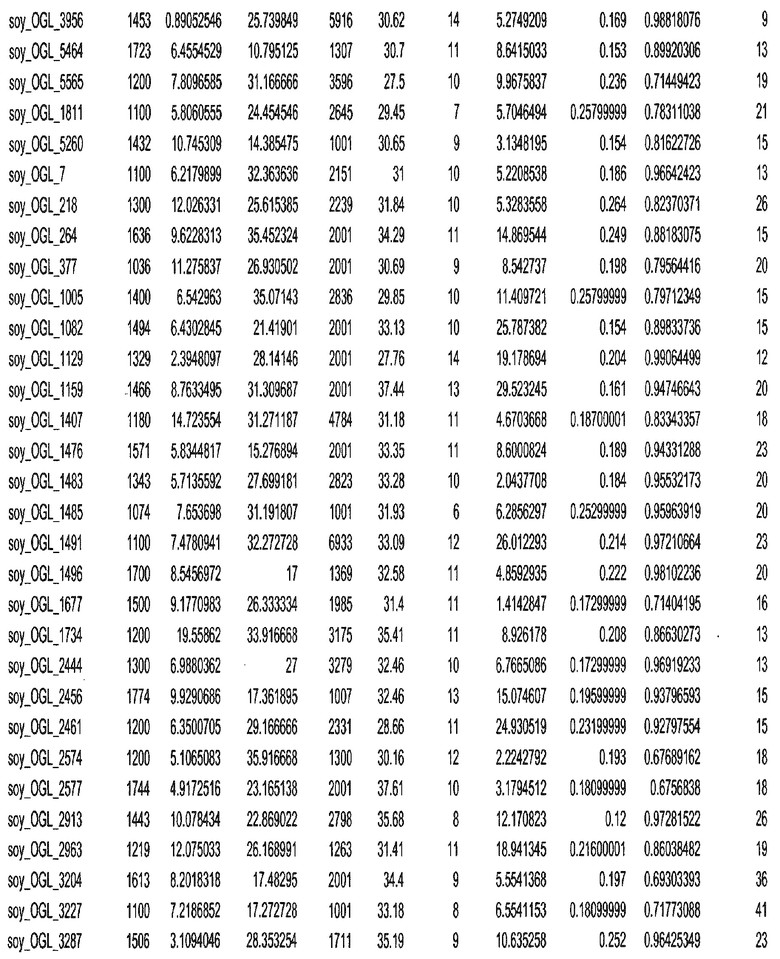
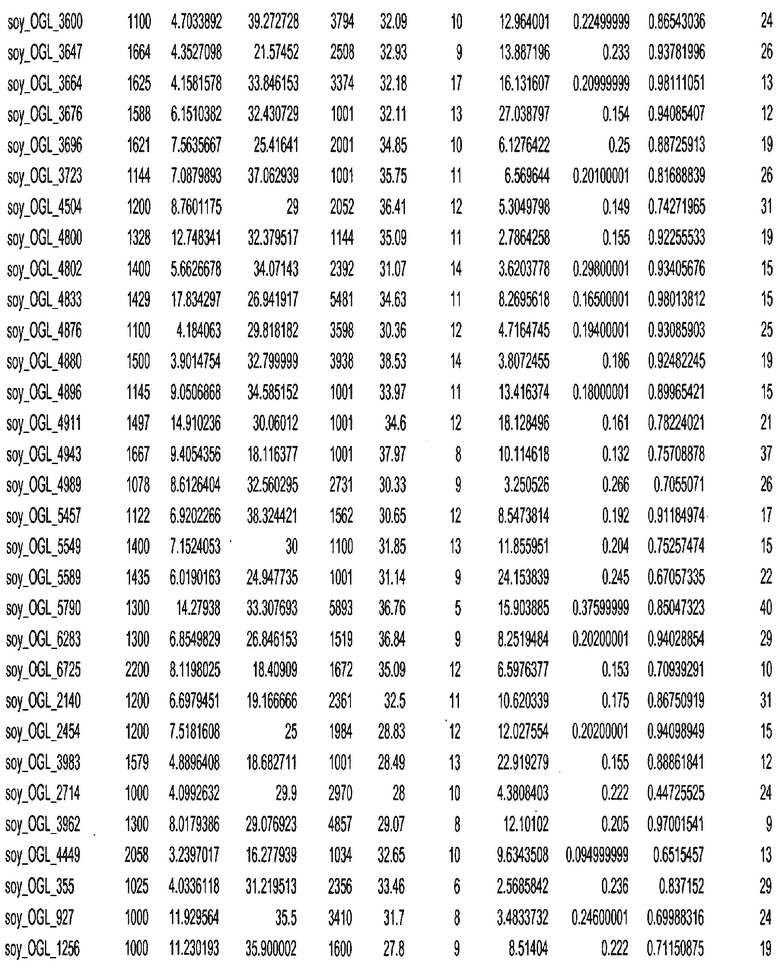
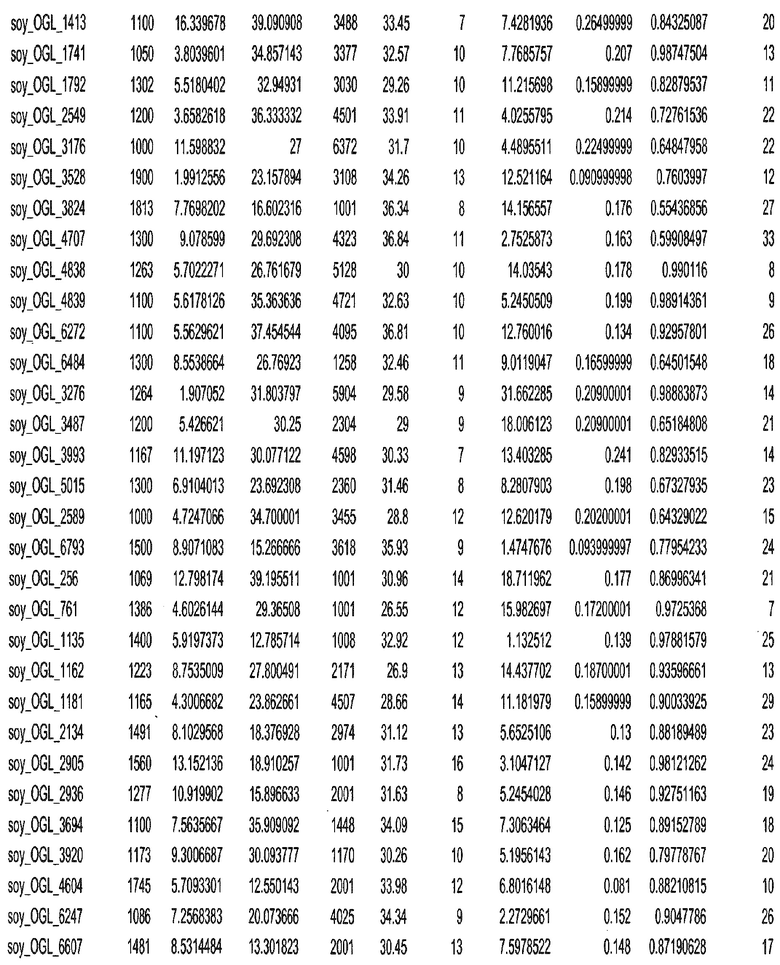
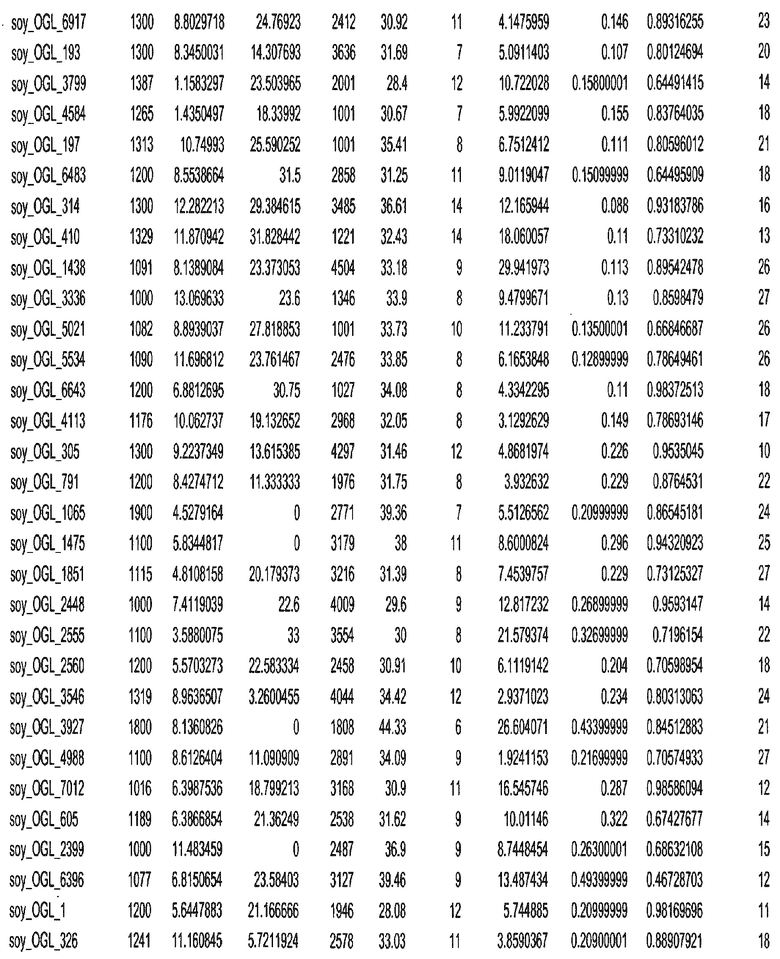
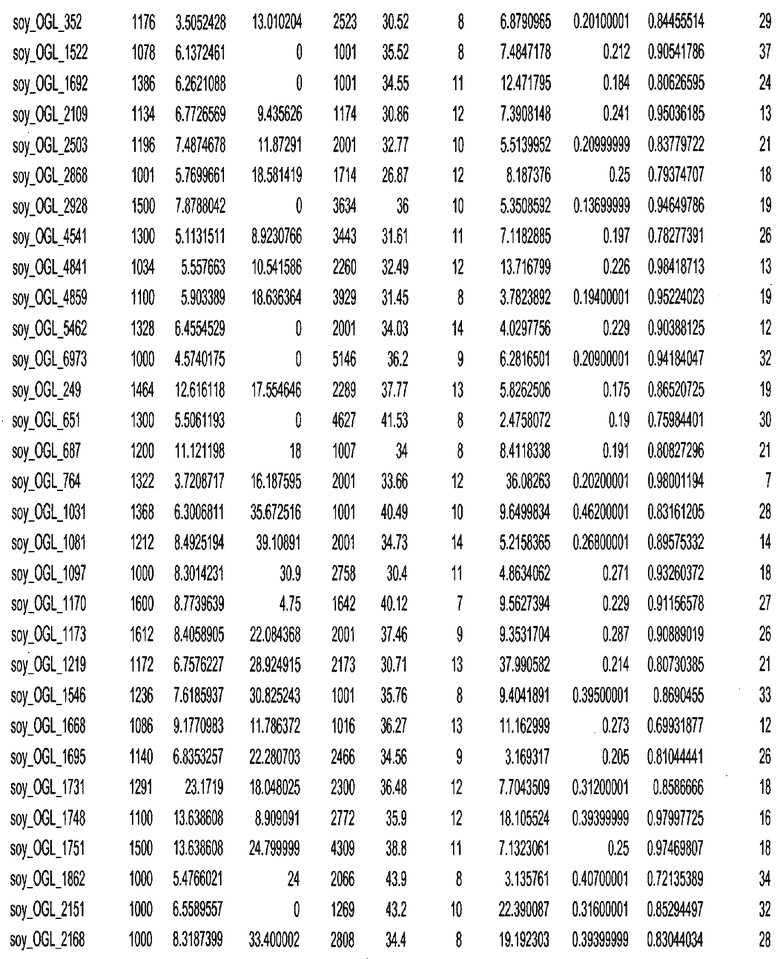
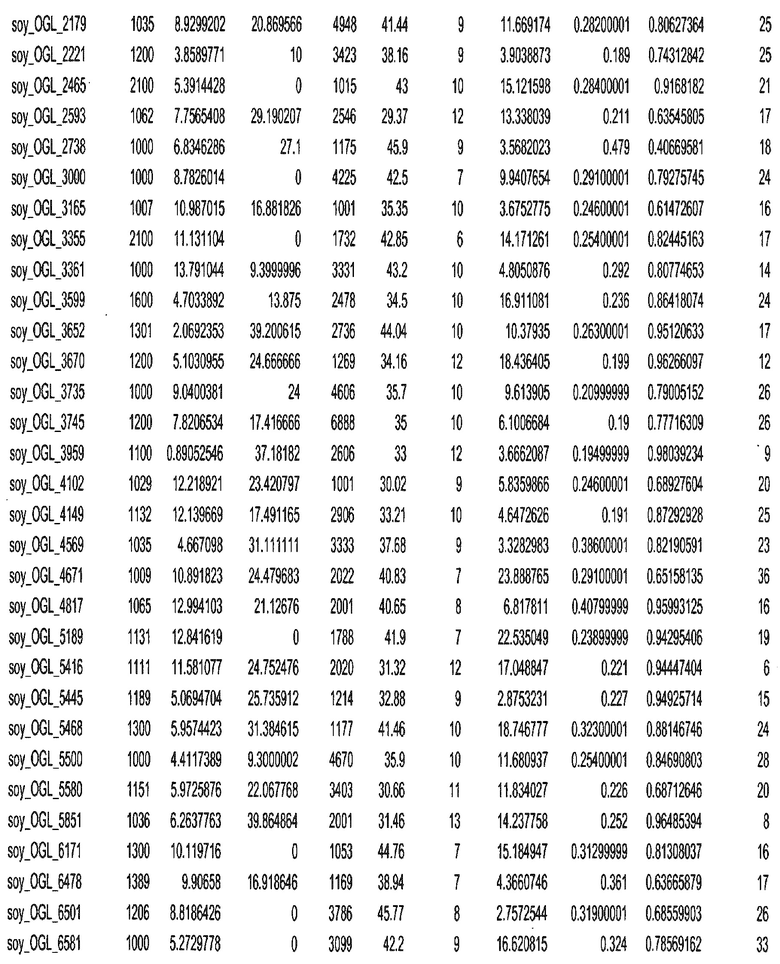
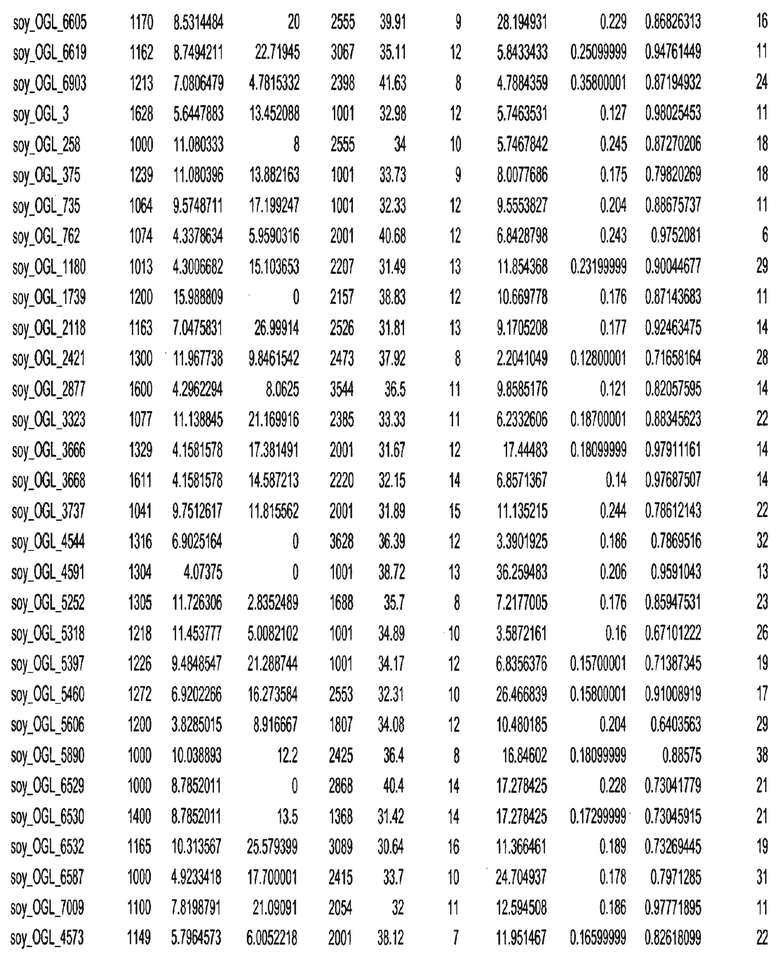

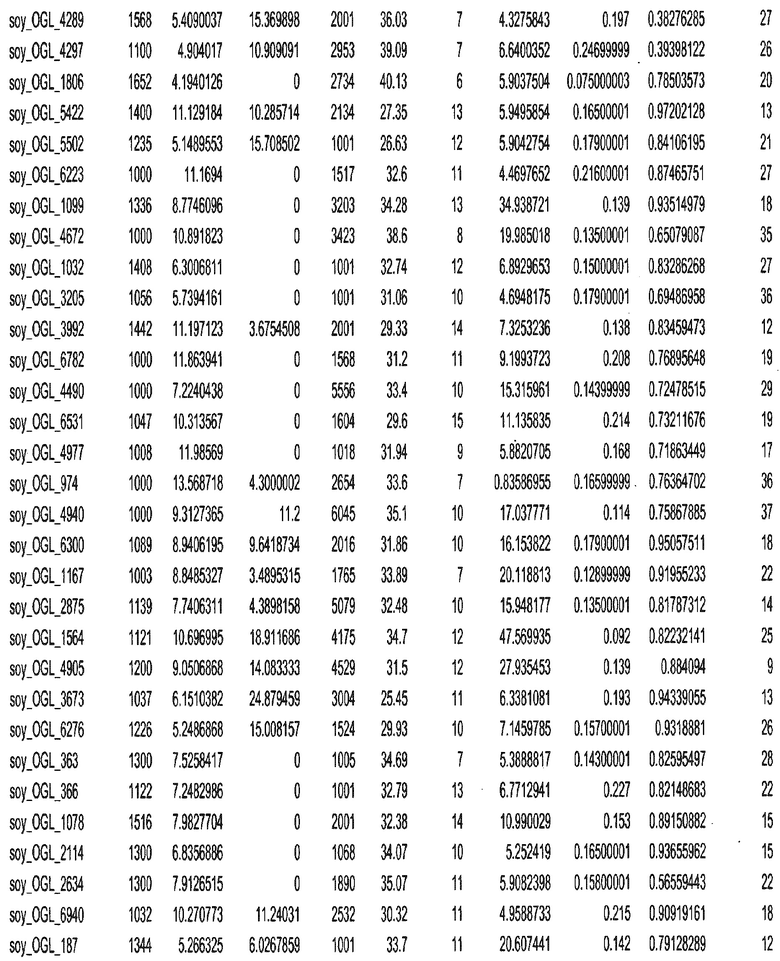
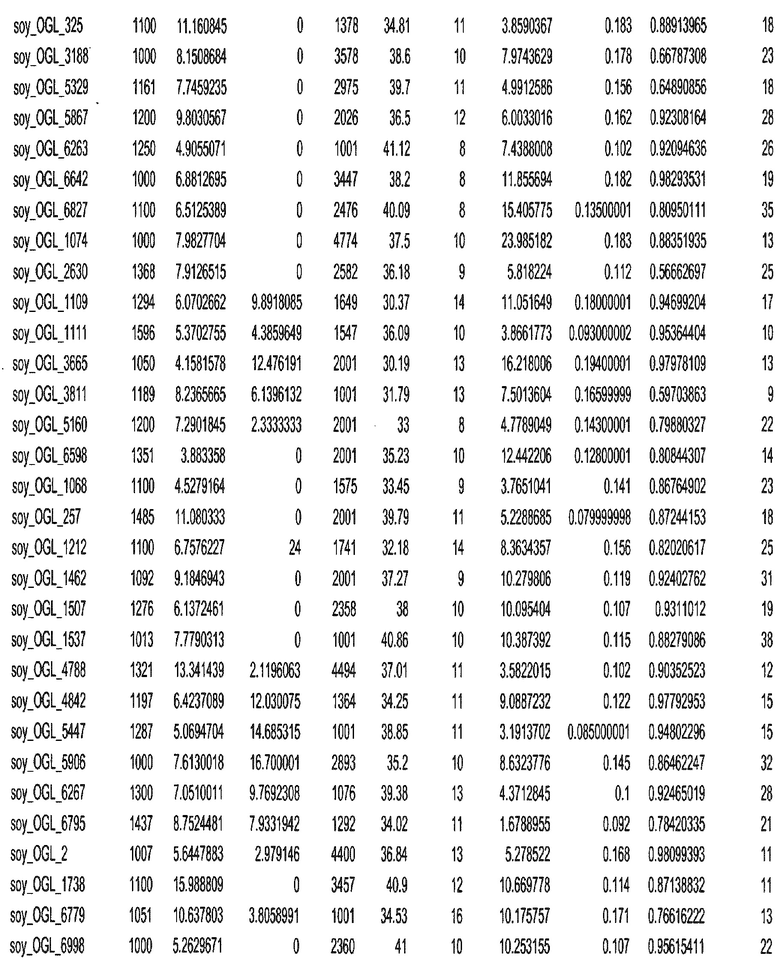
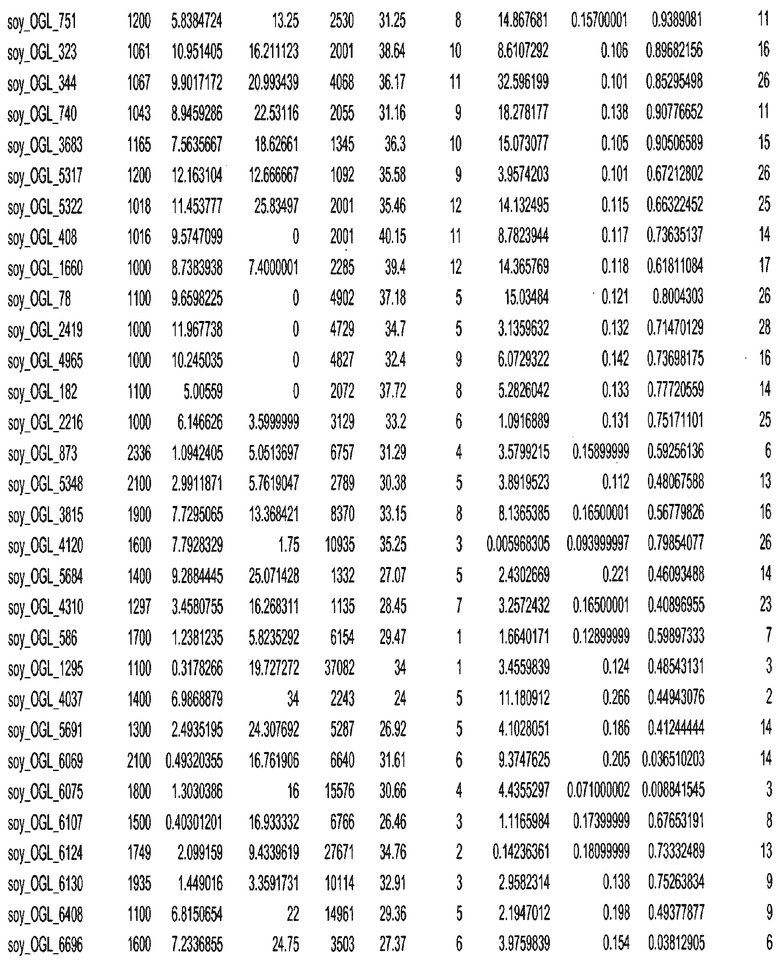
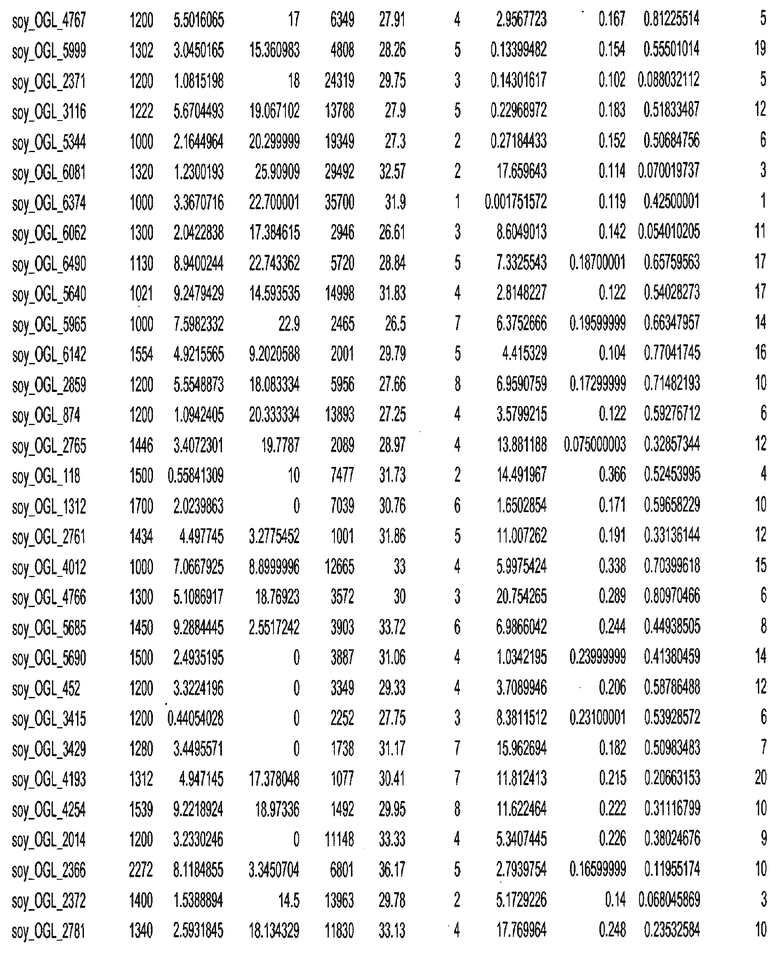
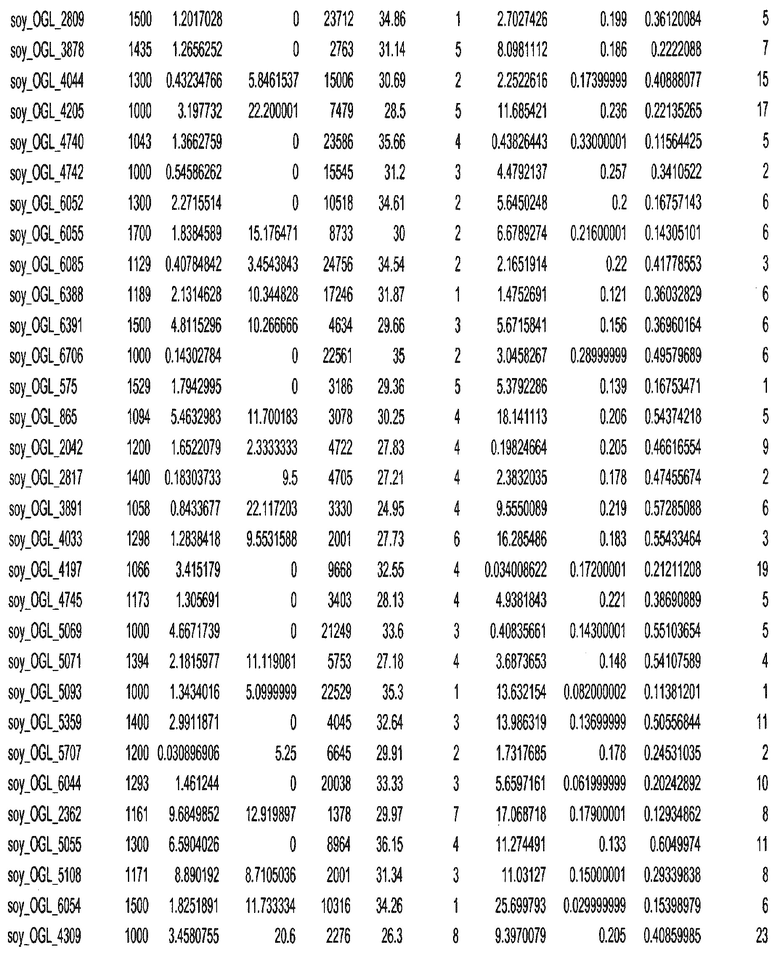
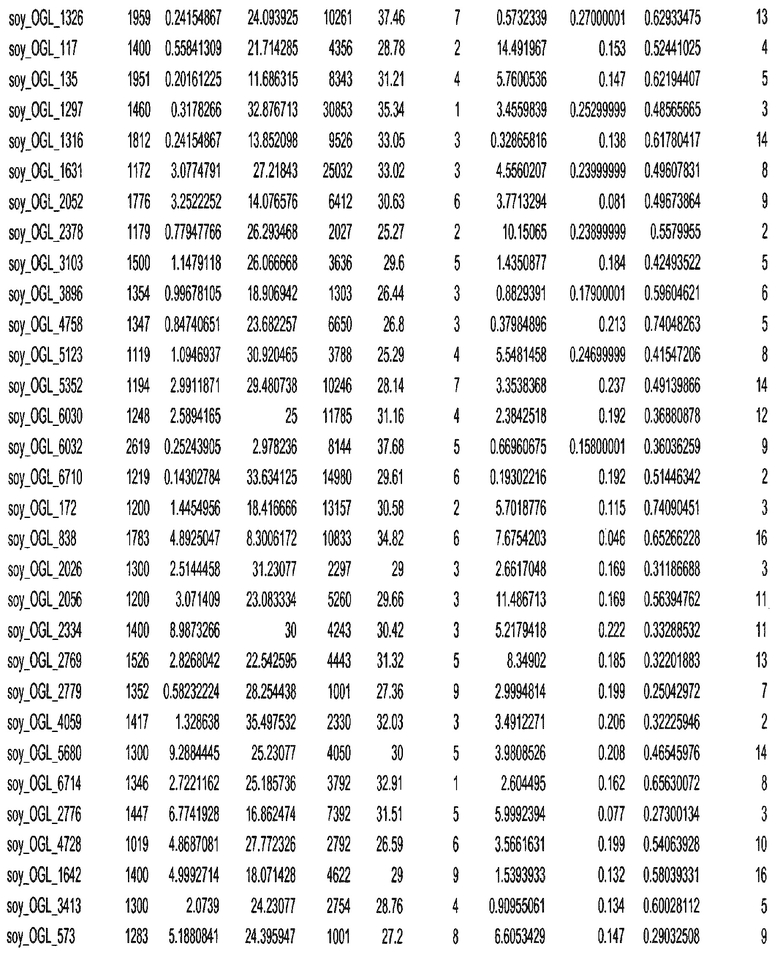
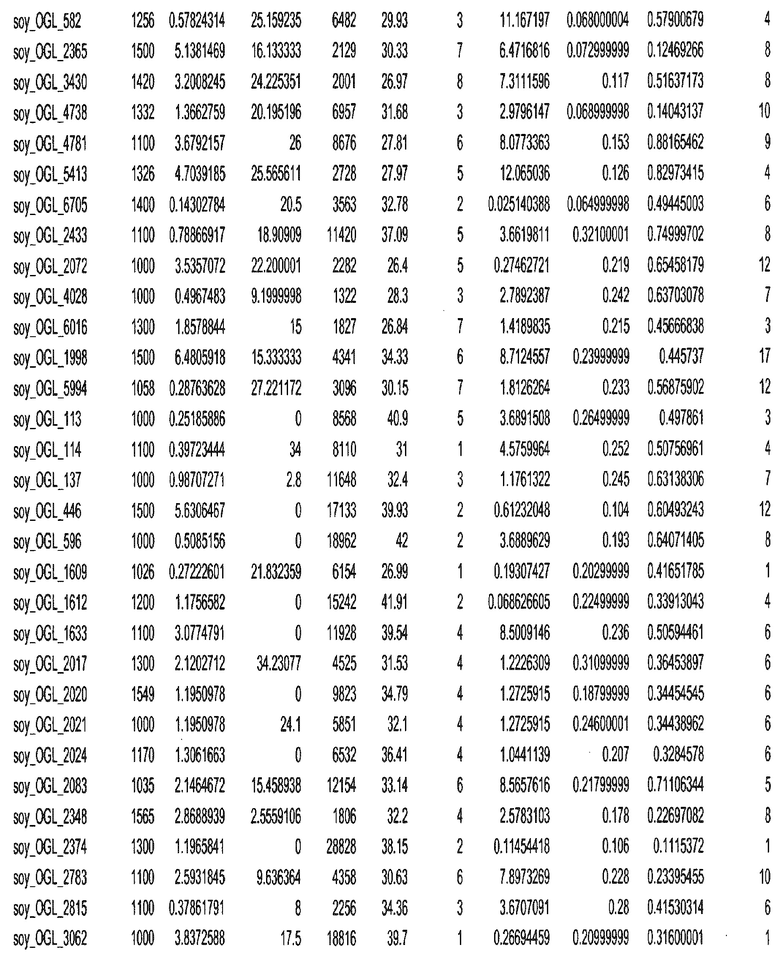
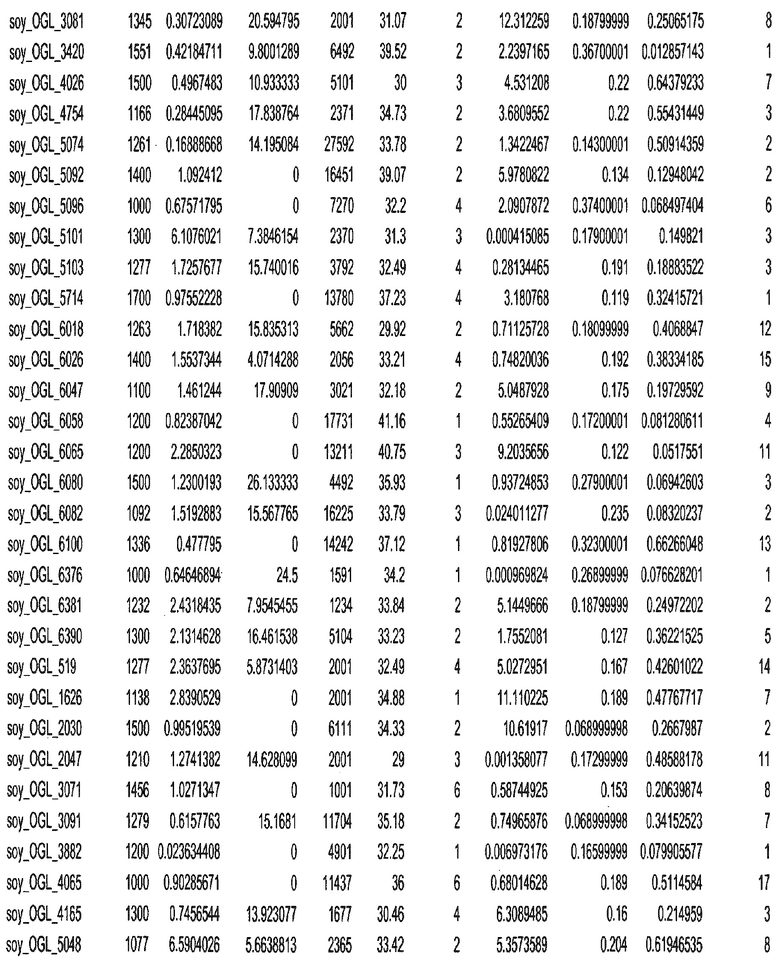
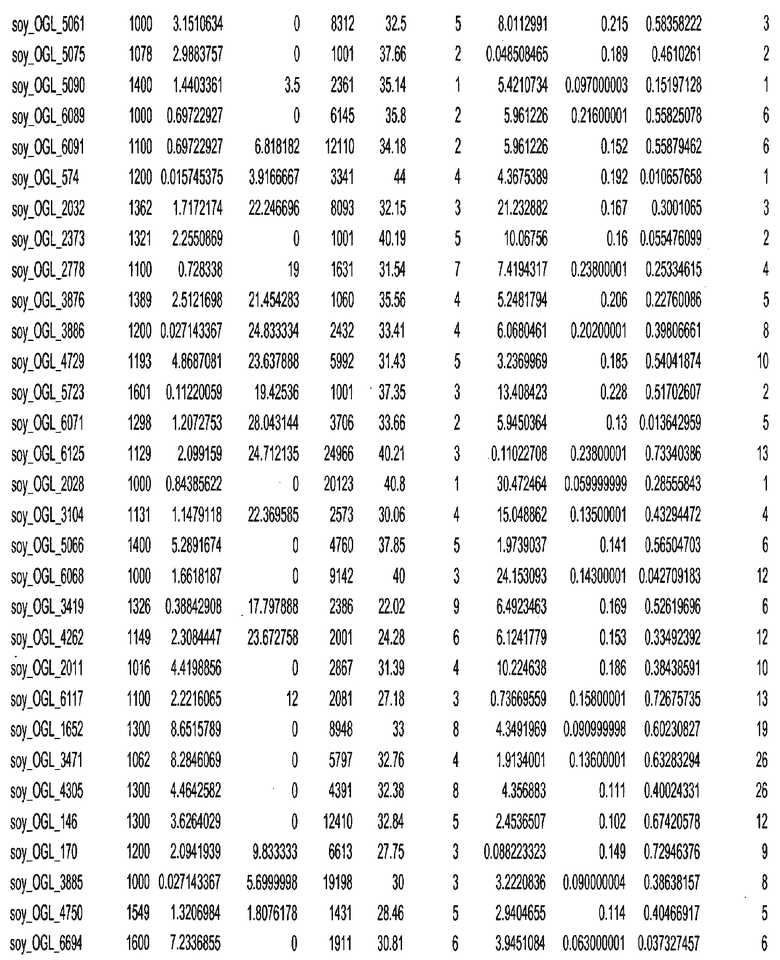
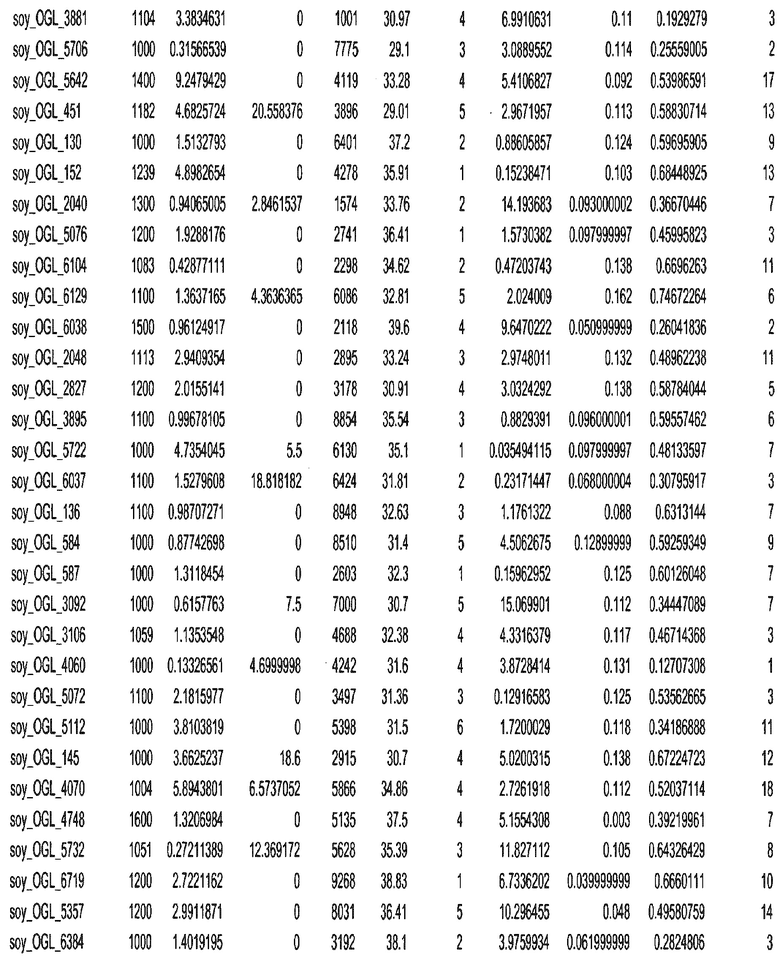
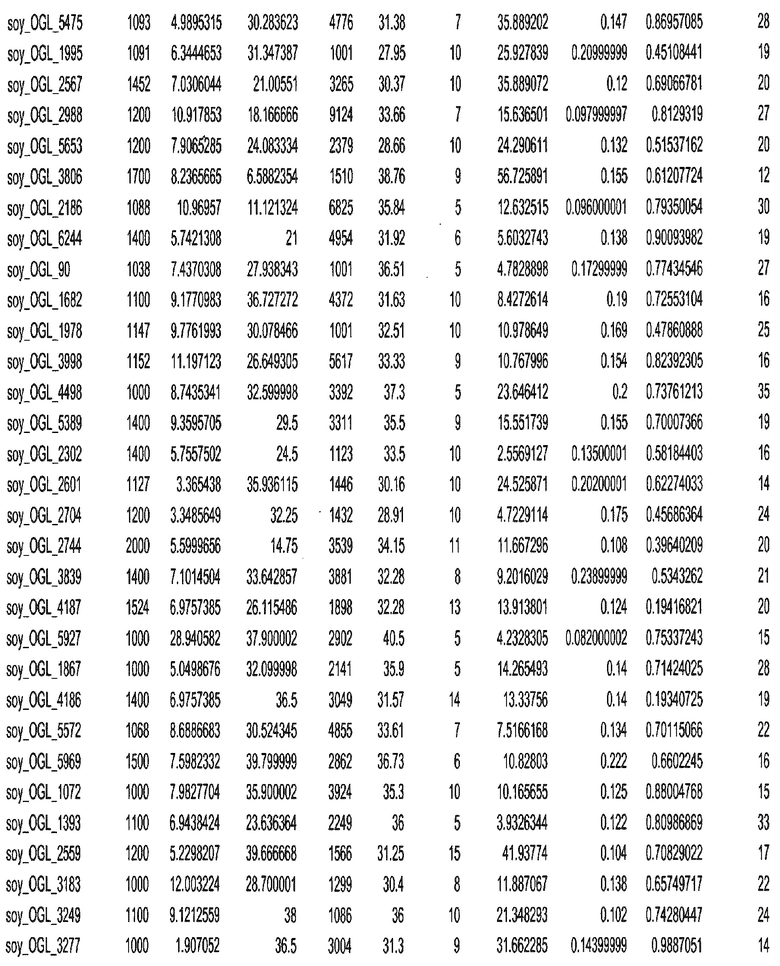
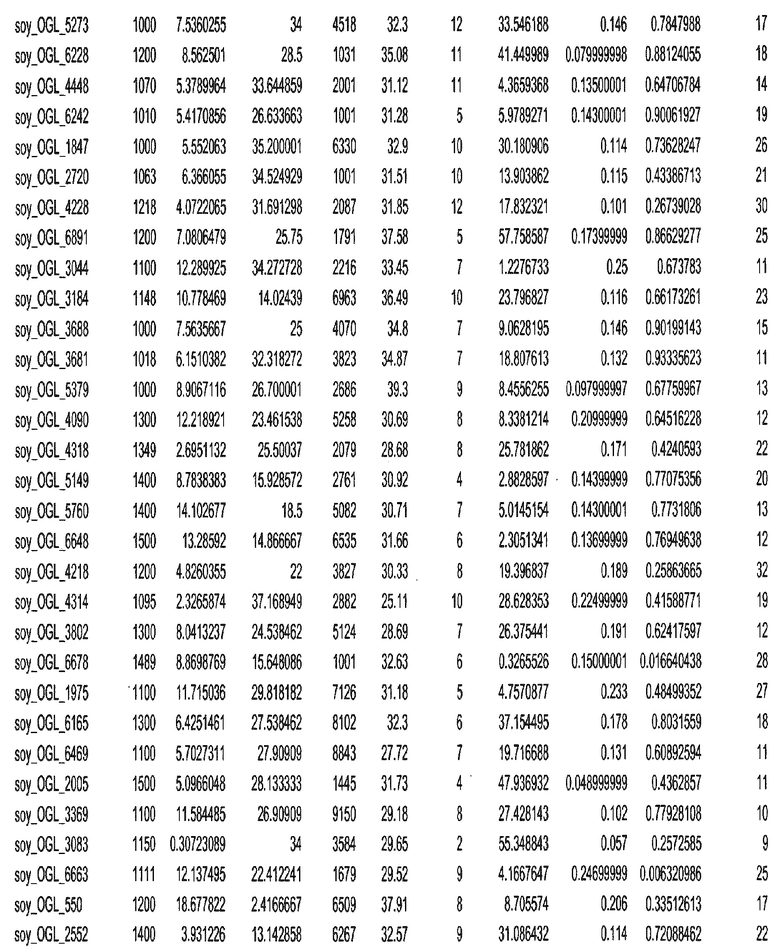
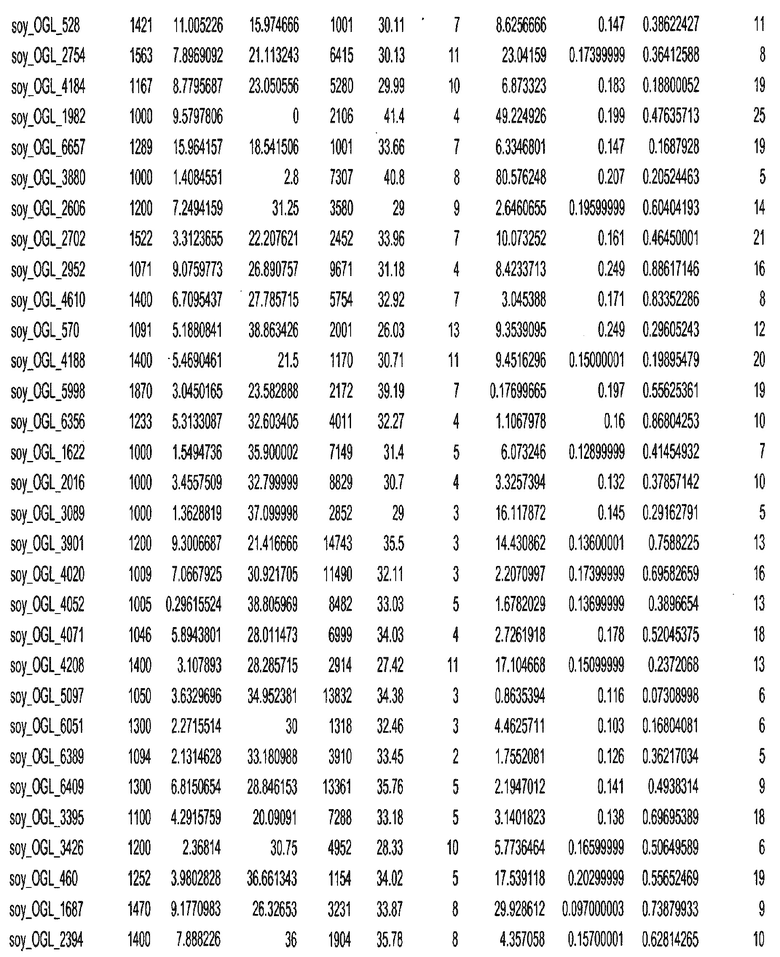
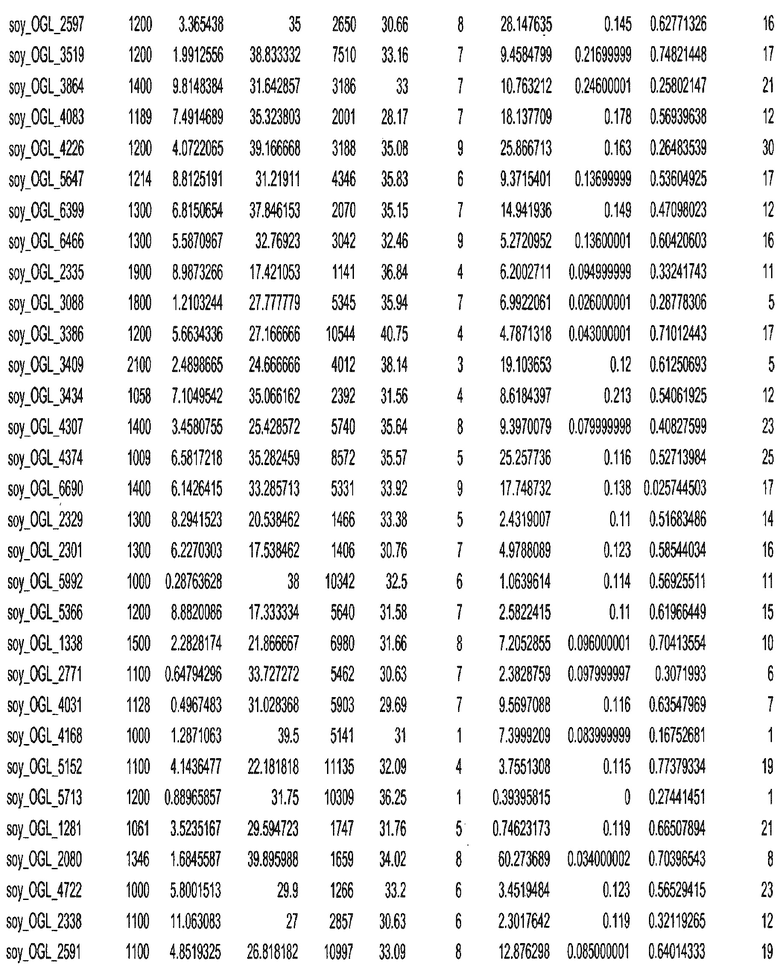
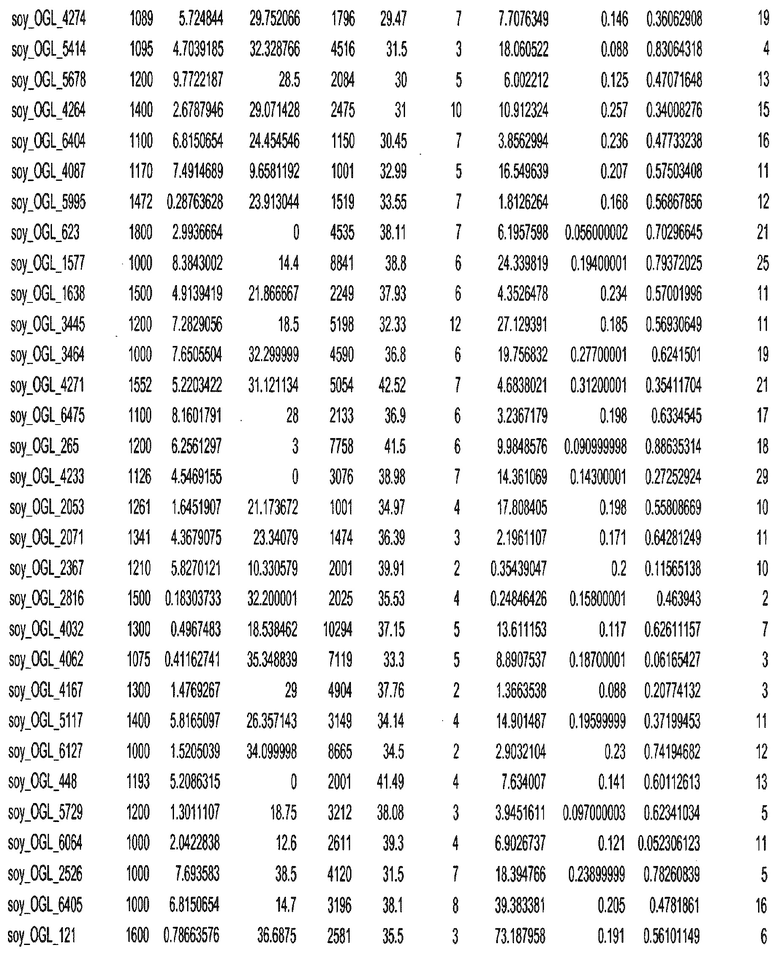
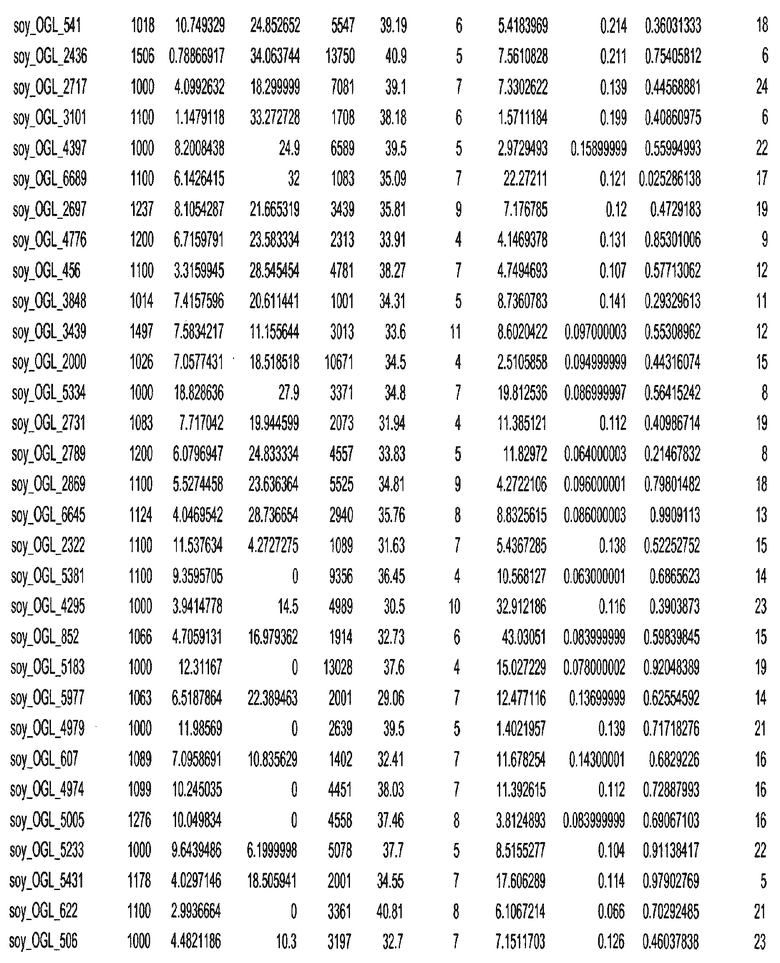
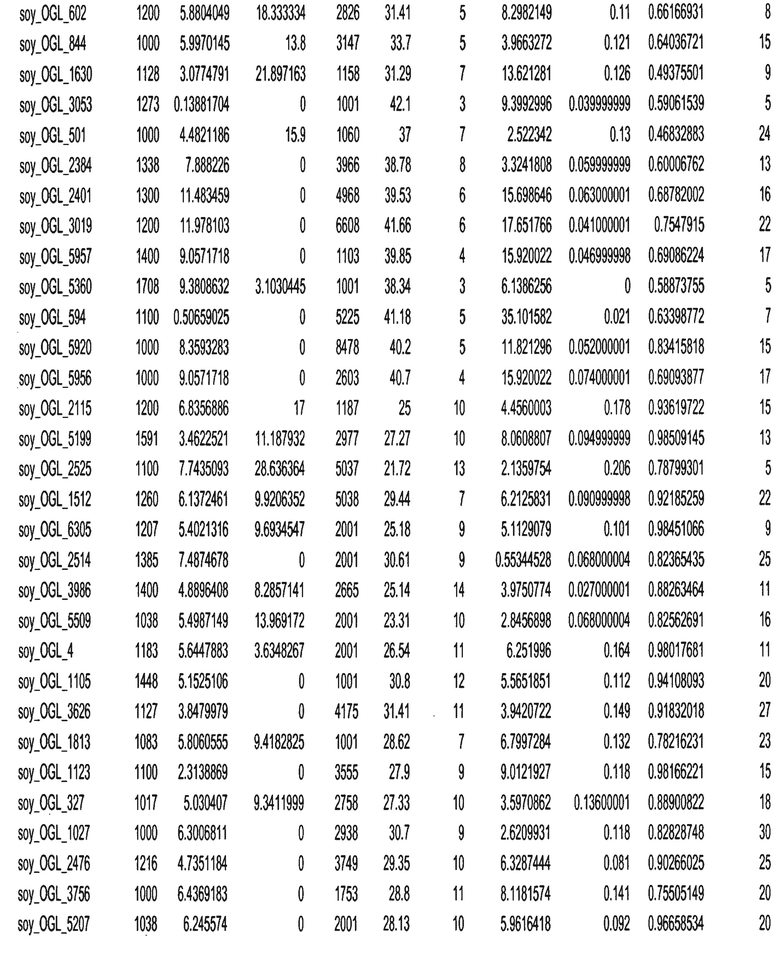
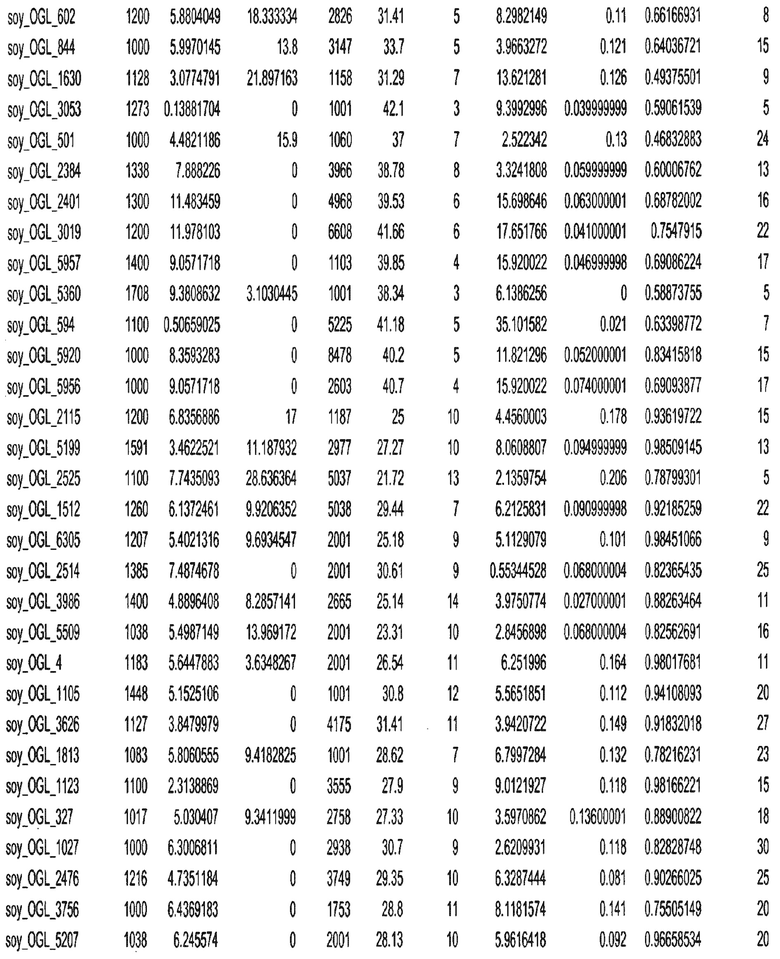
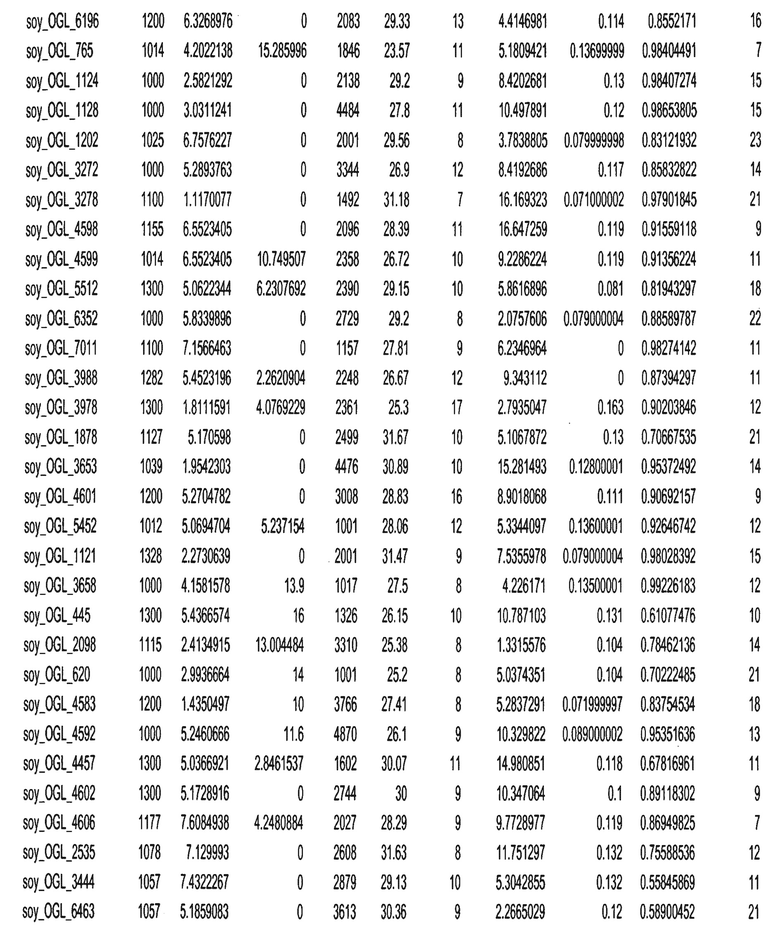
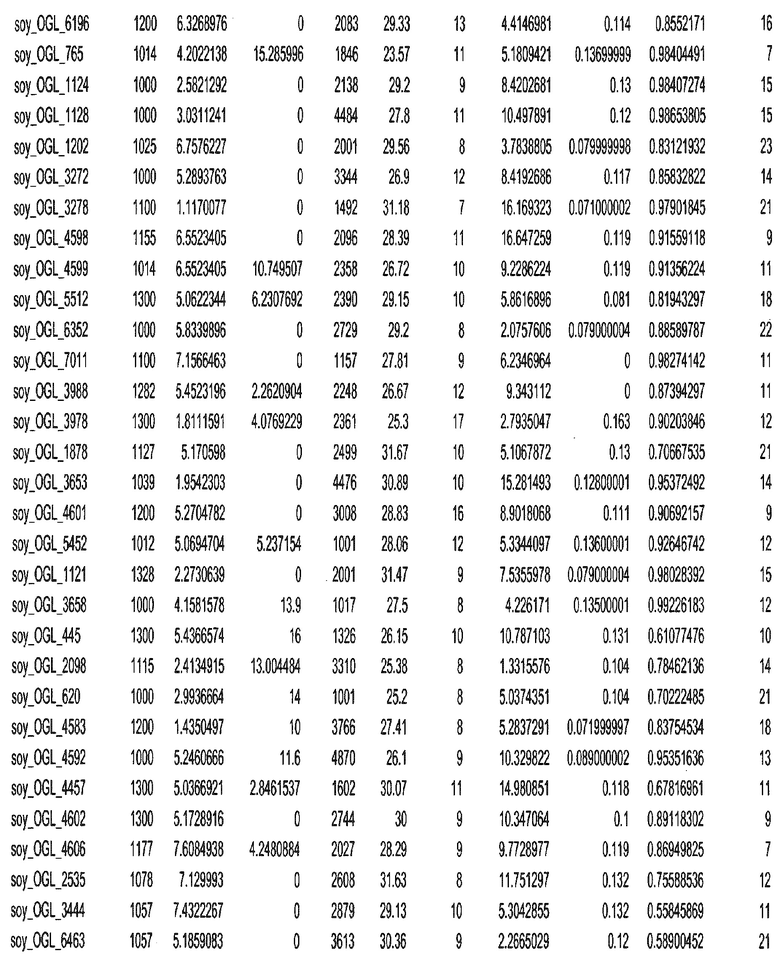
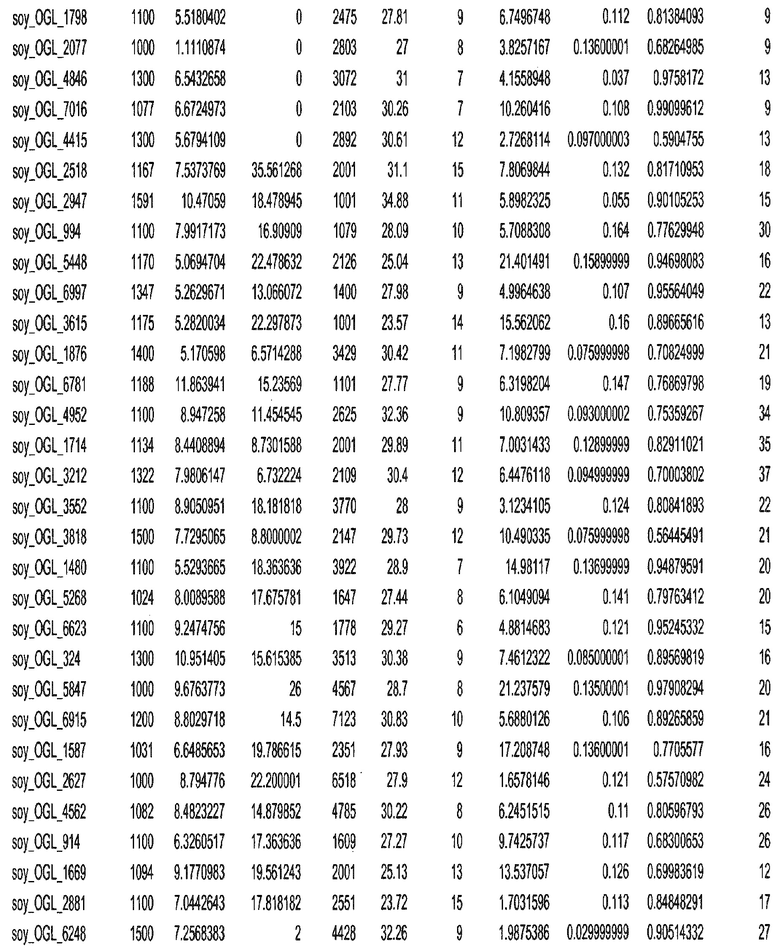
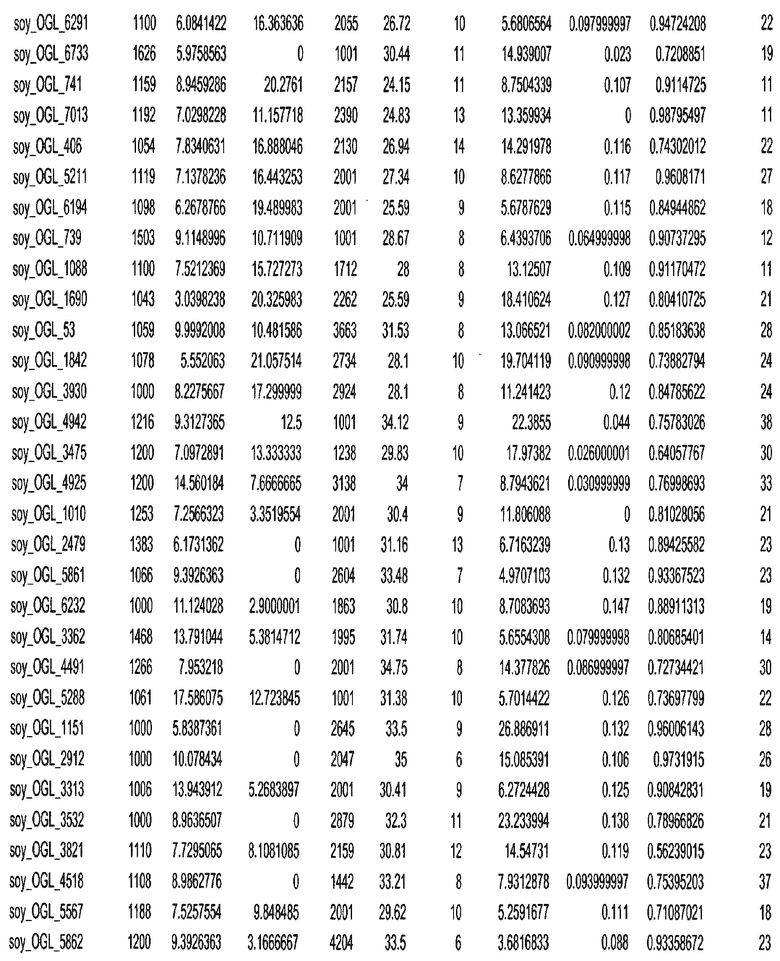
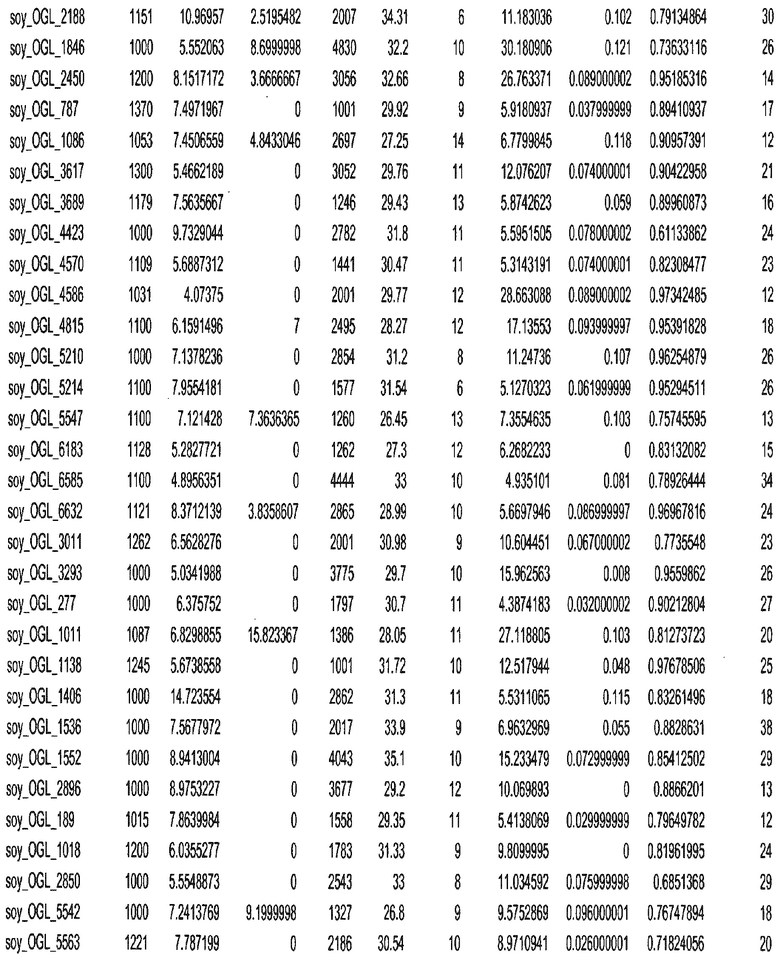
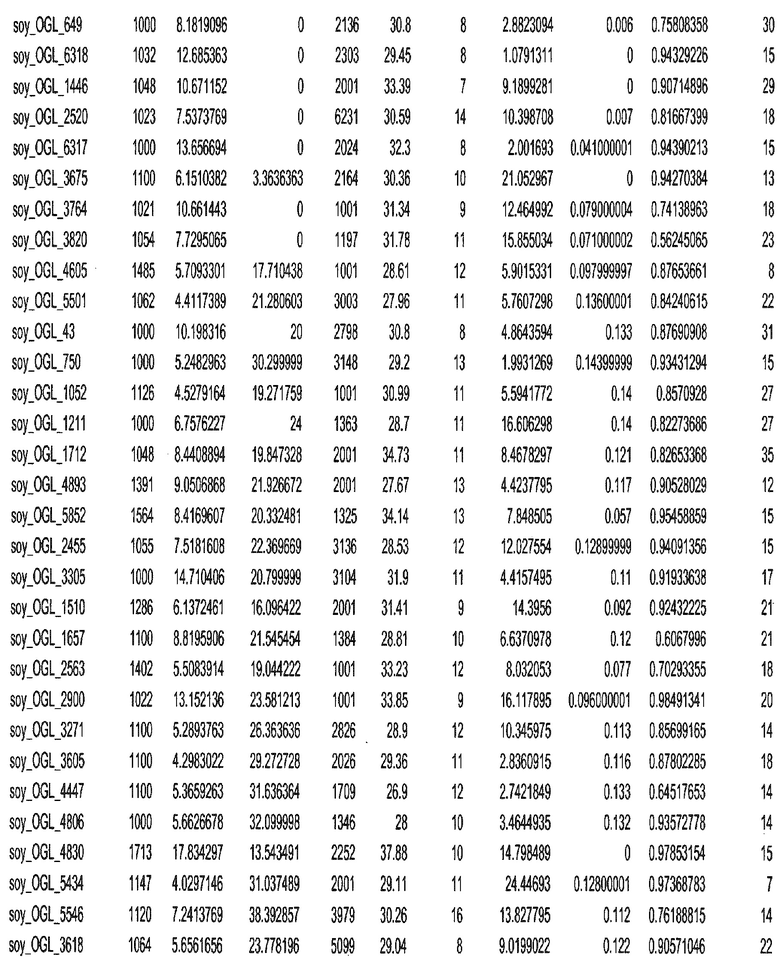
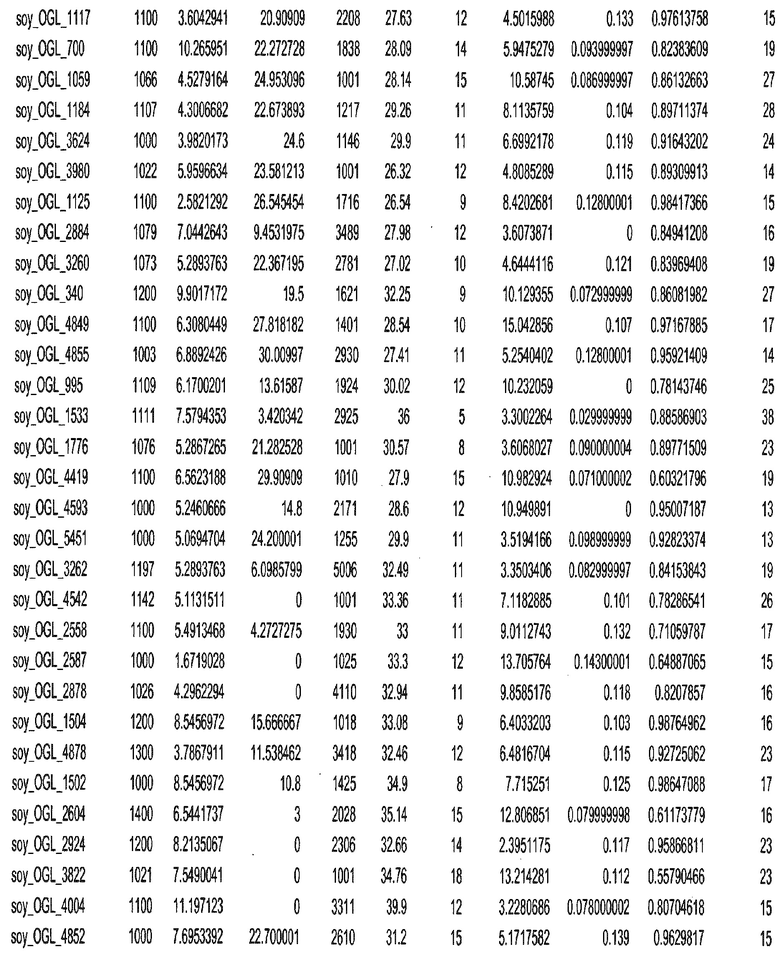
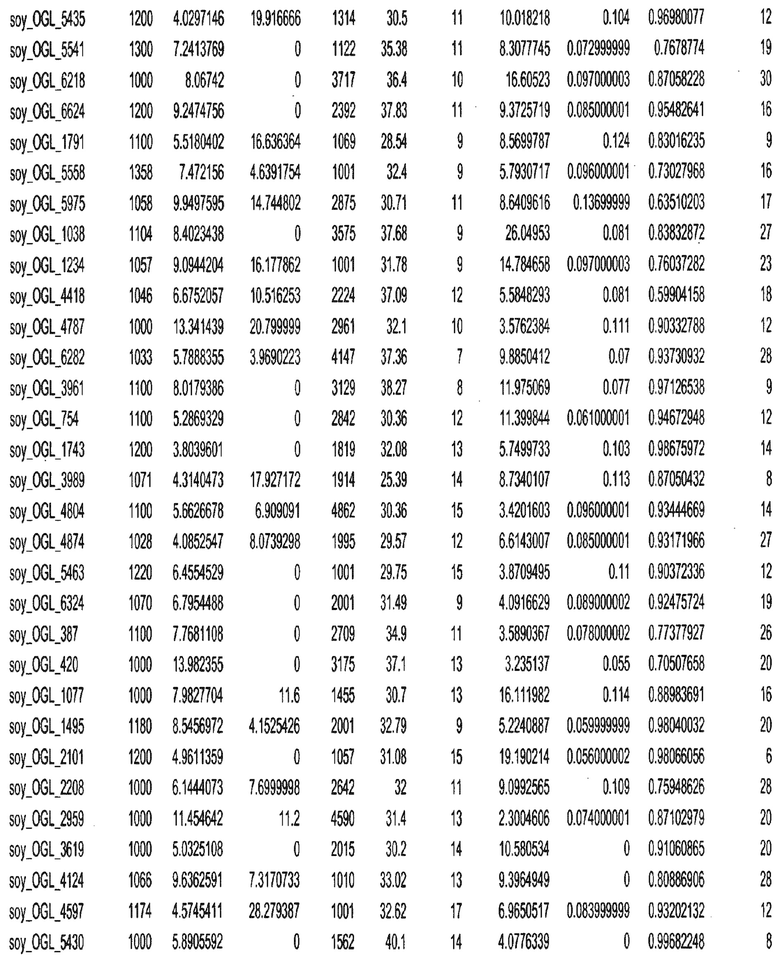
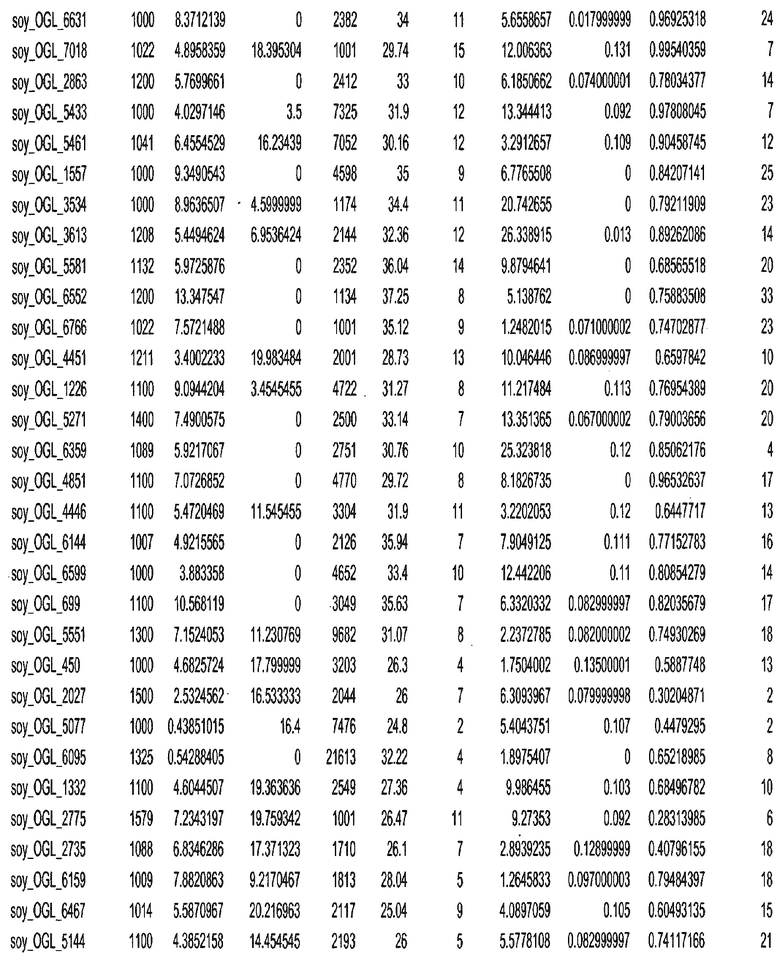
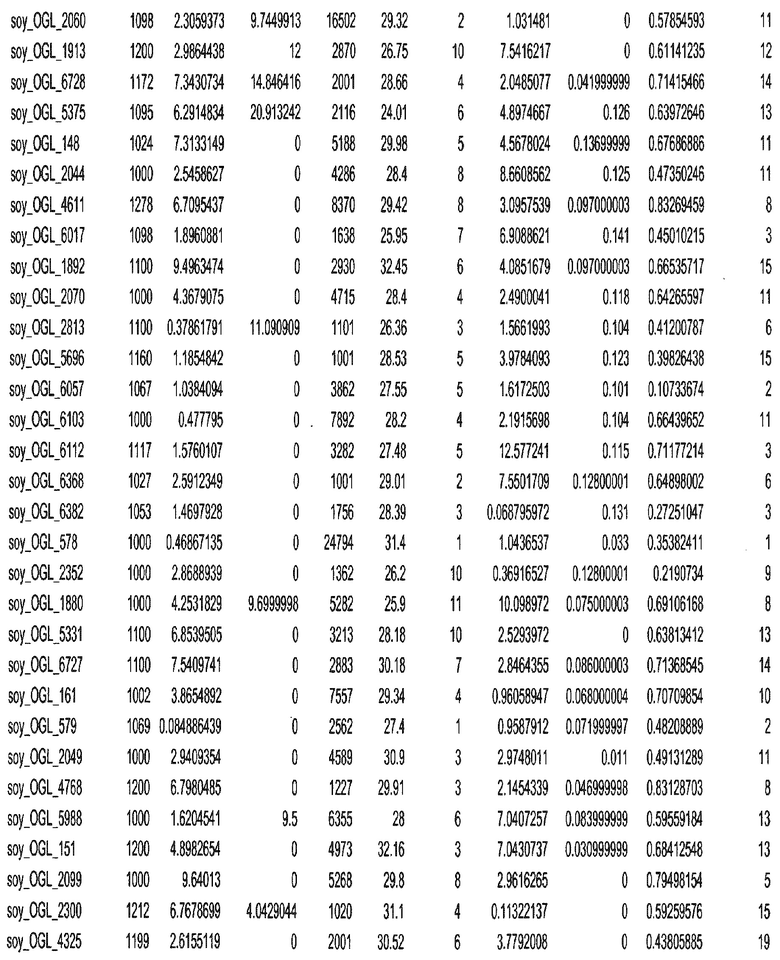
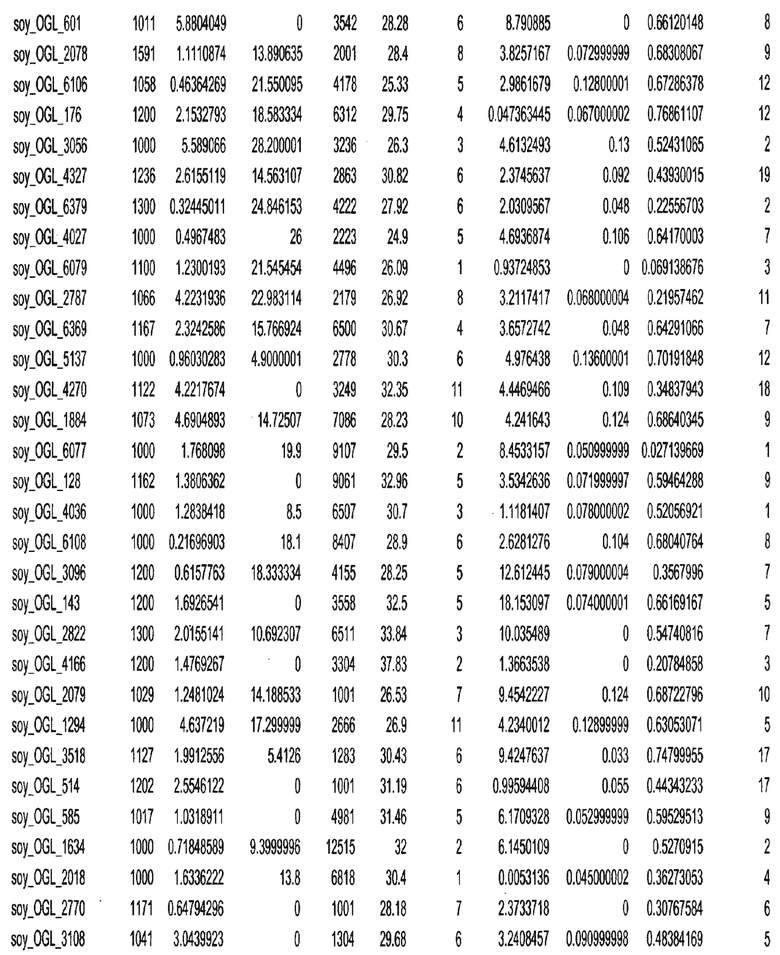
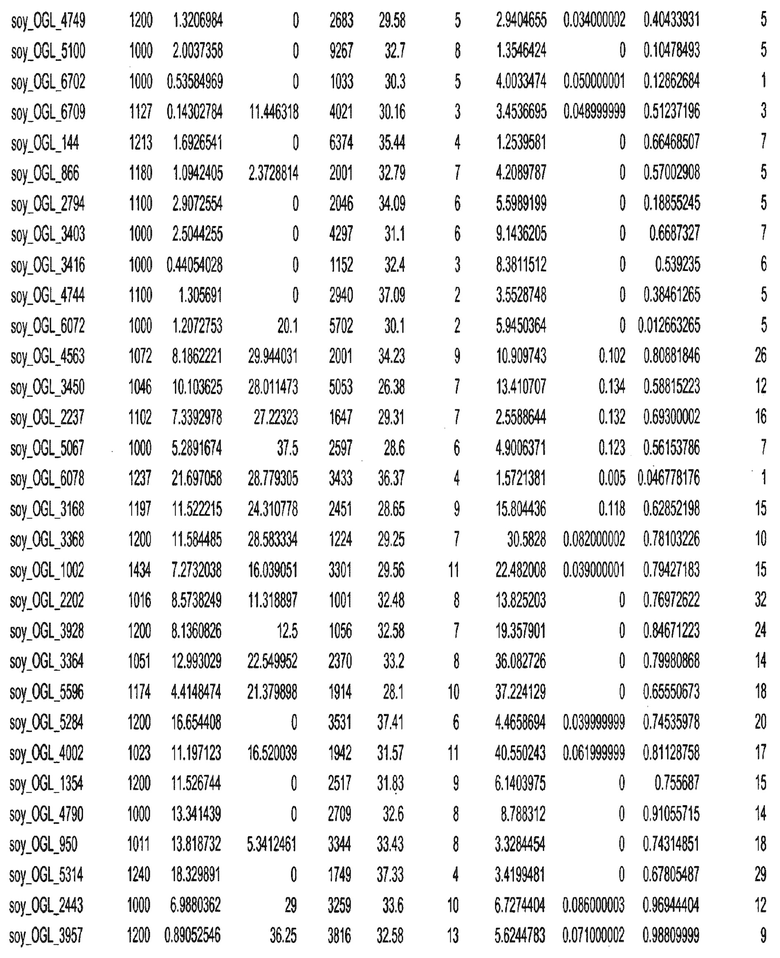
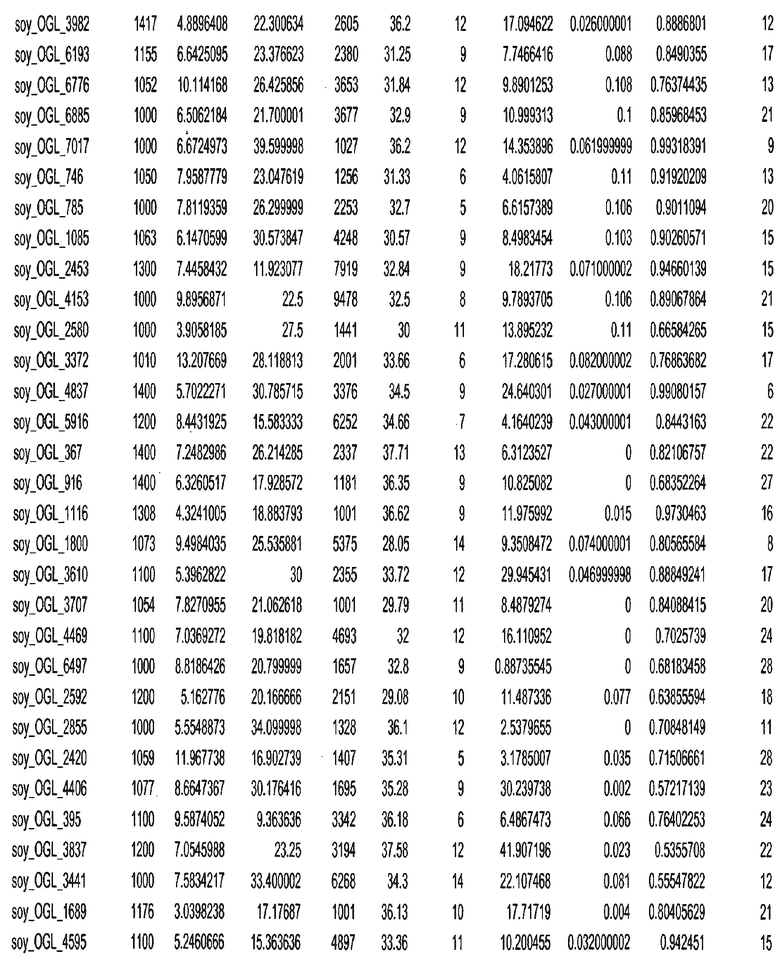
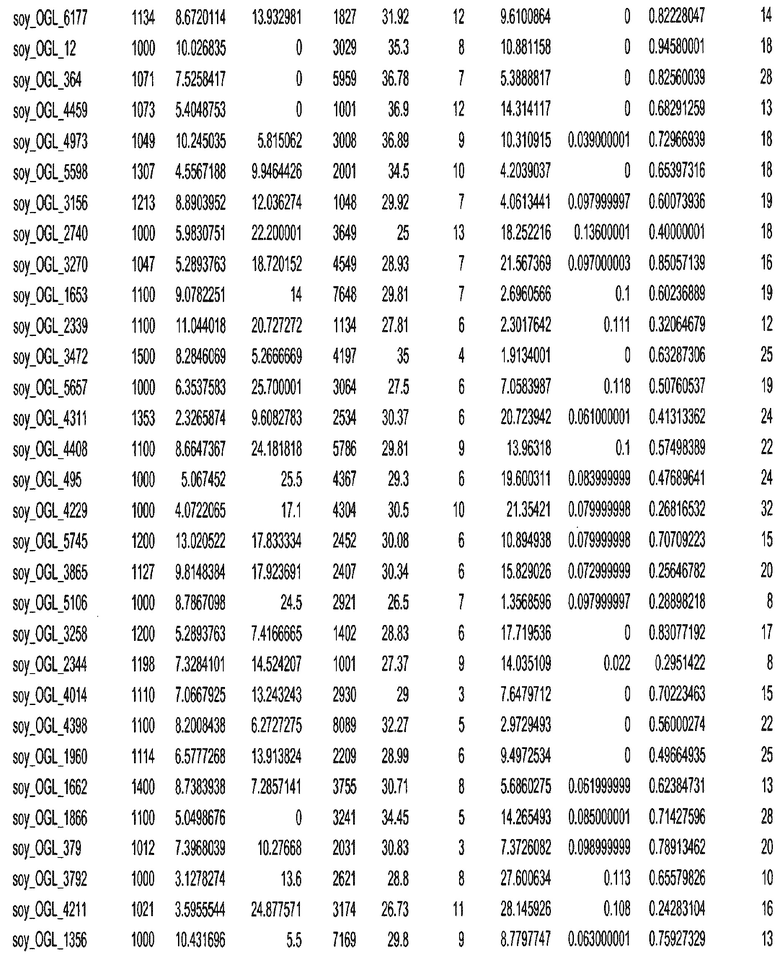
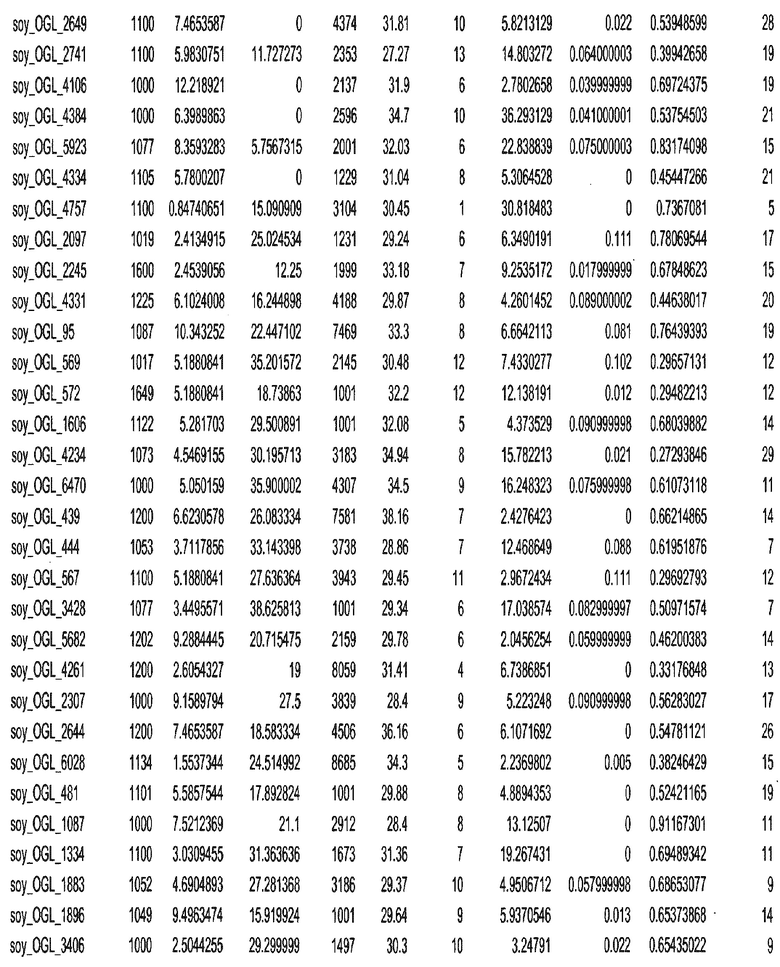
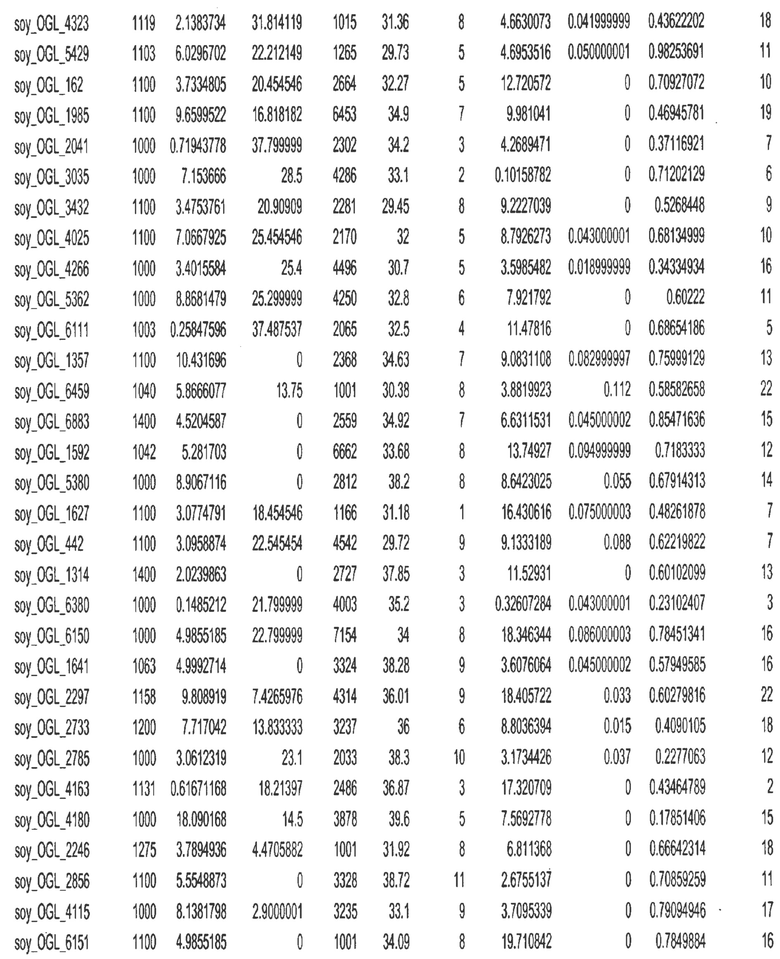
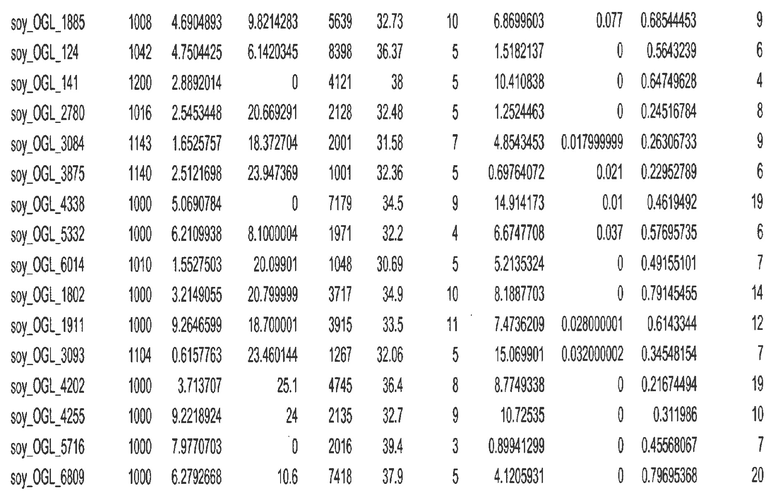
| название | год | авторы | номер документа |
|---|---|---|---|
| УНИВЕРСАЛЬНАЯ ДОНОРНАЯ СИСТЕМА ДЛЯ НАПРАВЛЕННОГО ВОЗДЕЙСТВИЯ НА ГЕНЫ | 2014 |
|
RU2687369C2 |
| БЫСТРЫЙ НАПРАВЛЕННЫЙ АНАЛИЗ СЕЛЬСКОХОЗЯЙСТВЕННЫХ КУЛЬТУР ДЛЯ ОПРЕДЕЛЕНИЯ ДОНОРНОЙ ВСТАВКИ | 2014 |
|
RU2668819C2 |
| СКОНСТРУИРОВАННАЯ СПОСОБАМИ ИНЖЕНЕРИИ ПЛАТФОРМА ДЛЯ ВСТРАИВАНИЯ ТРАНСГЕНА (ETIP) ДЛЯ НАЦЕЛИВАНИЯ ГЕНОВ И СТЭКИНГА ПРИЗНАКОВ | 2013 |
|
RU2666916C2 |
| ЛОКУСЫ ФУНКЦИОНАЛЬНОСТИ FAD2 И СООТВЕТСТВУЮЩИЕ СПЕЦИФИЧНЫЕ К УЧАСТКУ-МИШЕНИ СВЯЗЫВАЮЩИЕ БЕЛКИ, СПОСОБНЫЕ ИНДУЦИРОВАТЬ НАПРАВЛЕННЫЕ РАЗРЫВЫ | 2013 |
|
RU2656159C2 |
| ЛОКУСЫ FAD3 ДЛЯ ВЫПОЛНЕНИЯ ОПЕРАЦИЙ И СООТВЕТСТВУЮЩИЕ СВЯЗЫВАЮЩИЕСЯ СО СПЕЦИФИЧЕСКИМИ САЙТАМИ-МИШЕНЯМИ БЕЛКИ, СПОСОБНЫЕ К ВЫЗОВУ НАПРАВЛЕННЫХ РАЗРЫВОВ | 2013 |
|
RU2665811C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ОПОСРЕДОВАННОЙ НУКЛЕАЗОЙ НАПРАВЛЕННОЙ ИНТЕГРАЦИИ ТРАНСГЕНОВ | 2013 |
|
RU2650819C2 |
| ОБОГАЩЕНИЕ АКТИВИРУЕМОЙ ФЛУОРЕСЦЕНЦИЕЙ СОРТИРОВКИ КЛЕТОК (FACS) ДЛЯ СОЗДАНИЯ РАСТЕНИЙ | 2013 |
|
RU2679510C2 |
| ФУНКЦИОНАЛЬНЫЕ ЛОКУСЫ FAD2 И СООТВЕТСТВУЮЩИЕ СПЕЦИФИЧНЫЕ ДЛЯ САЙТА-МИШЕНИ СВЯЗЫВАЮЩИЕСЯ БЕЛКИ, СПОСОБНЫЕ ИНДУЦИРОВАТЬ НАПРАВЛЕННЫЕ РАЗРЫВЫ | 2013 |
|
RU2656158C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ВСТРАИВАНИЯ ЭКЗОГЕННОЙ ПОСЛЕДОВАТЕЛЬНОСТИ В ГЕНОМ РАСТЕНИЙ | 2014 |
|
RU2723130C2 |
| ГЕН И ВАРИАЦИИ, СВЯЗАННЫЕ С ФЕНОТИПОМ BM1, МОЛЕКУЛЯРНЫЕ МАРКЕРЫ И ИХ ПРИМЕНЕНИЕ | 2012 |
|
RU2617958C2 |
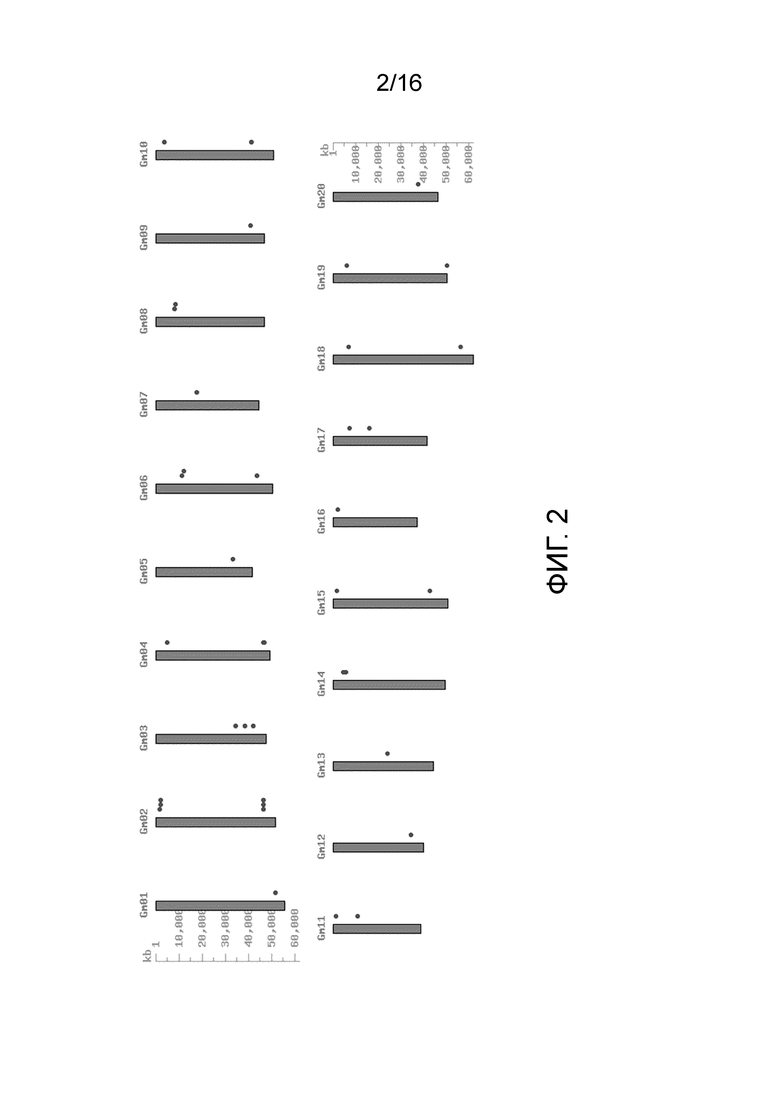

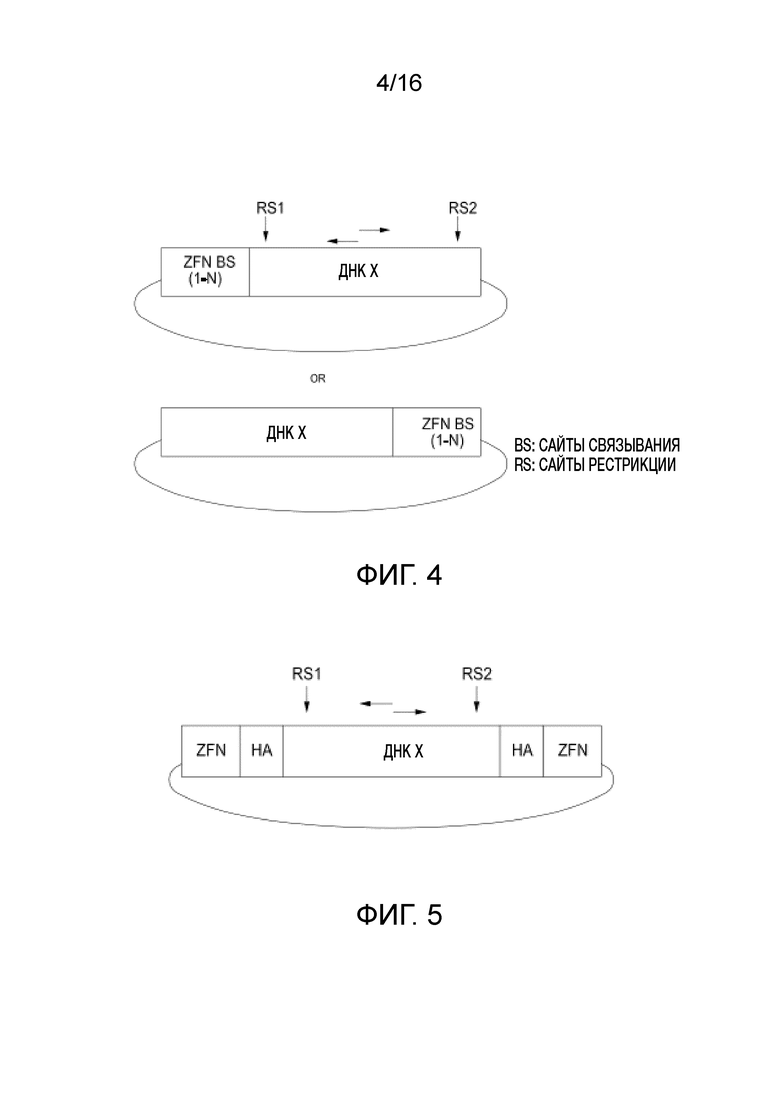
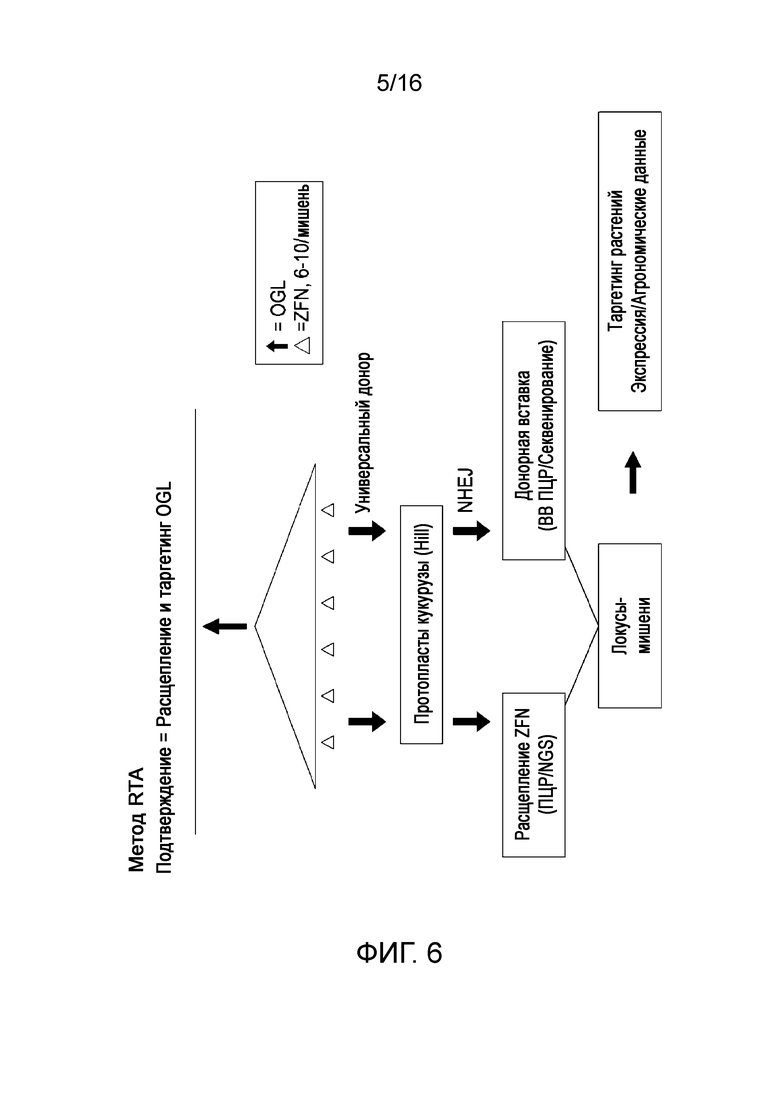
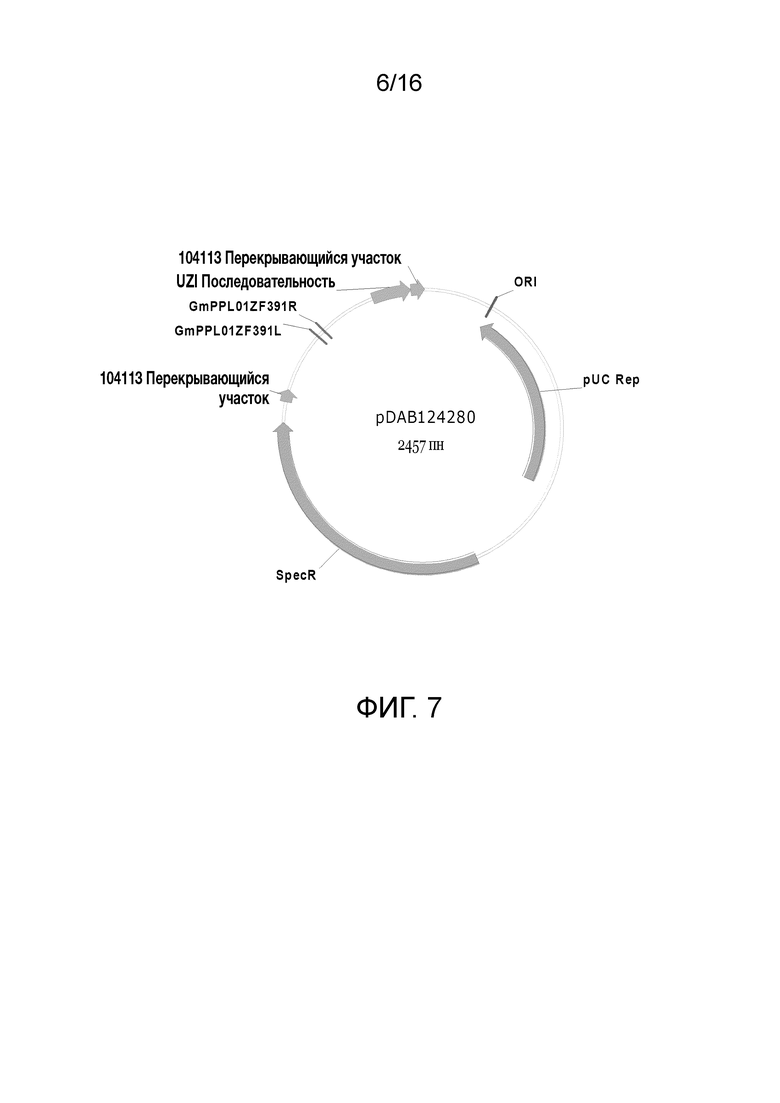
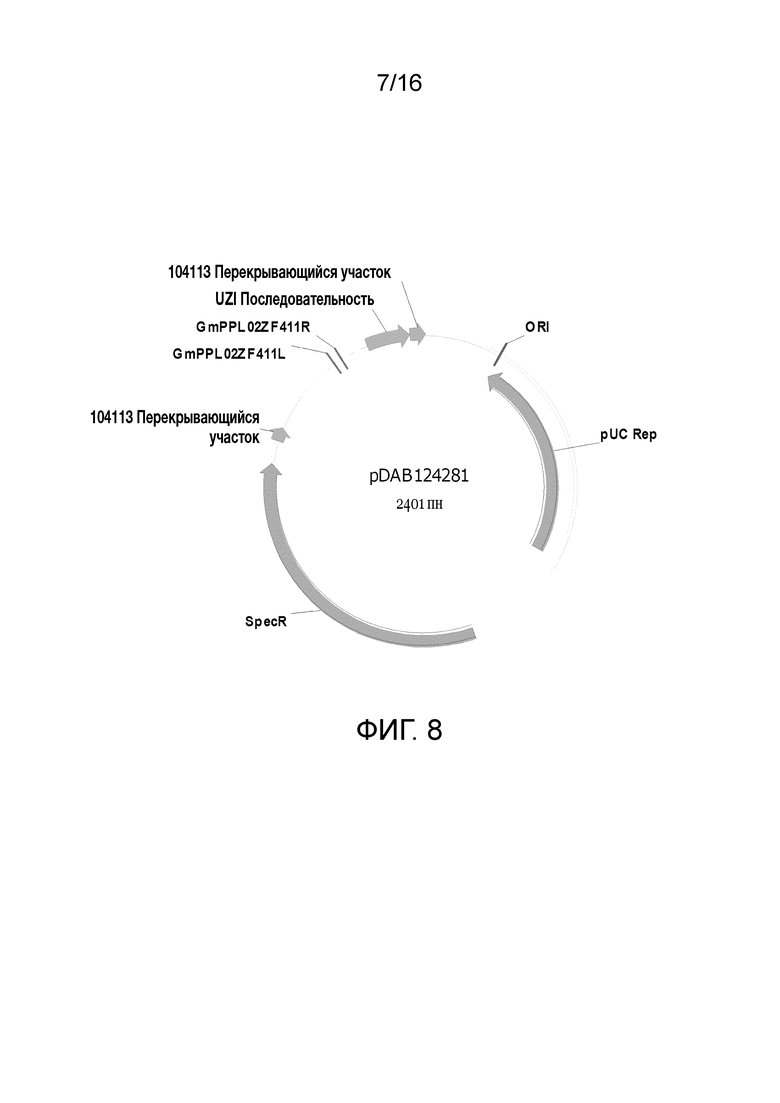
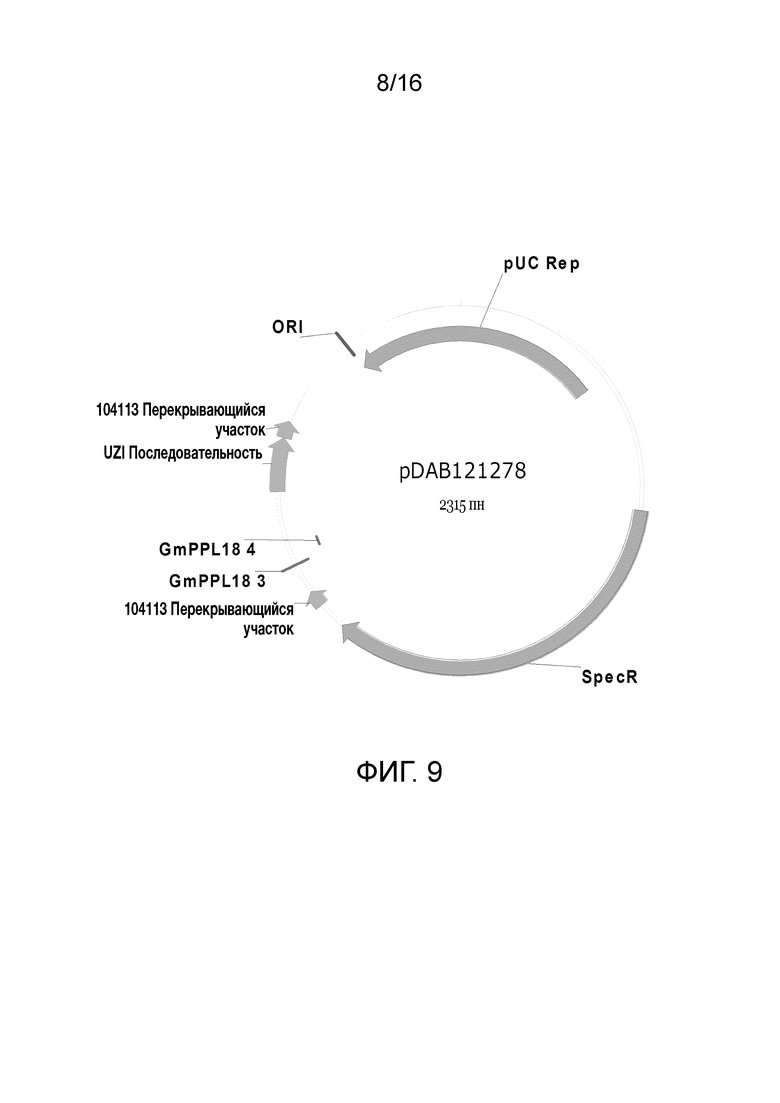
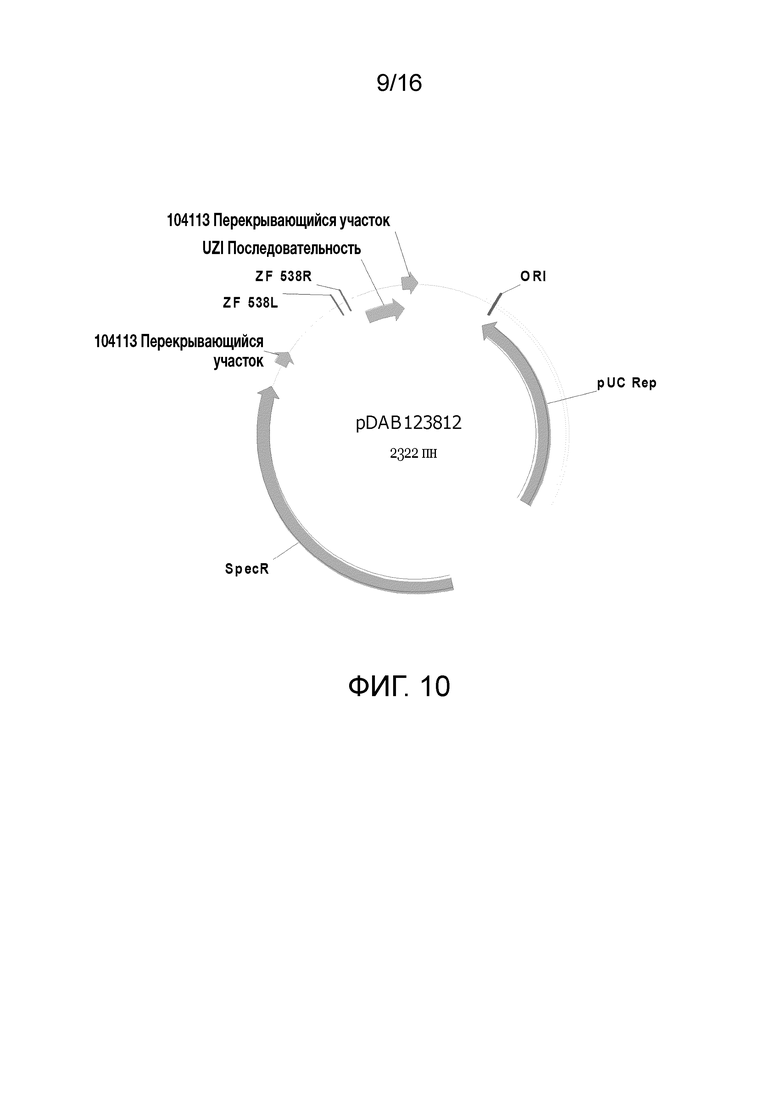
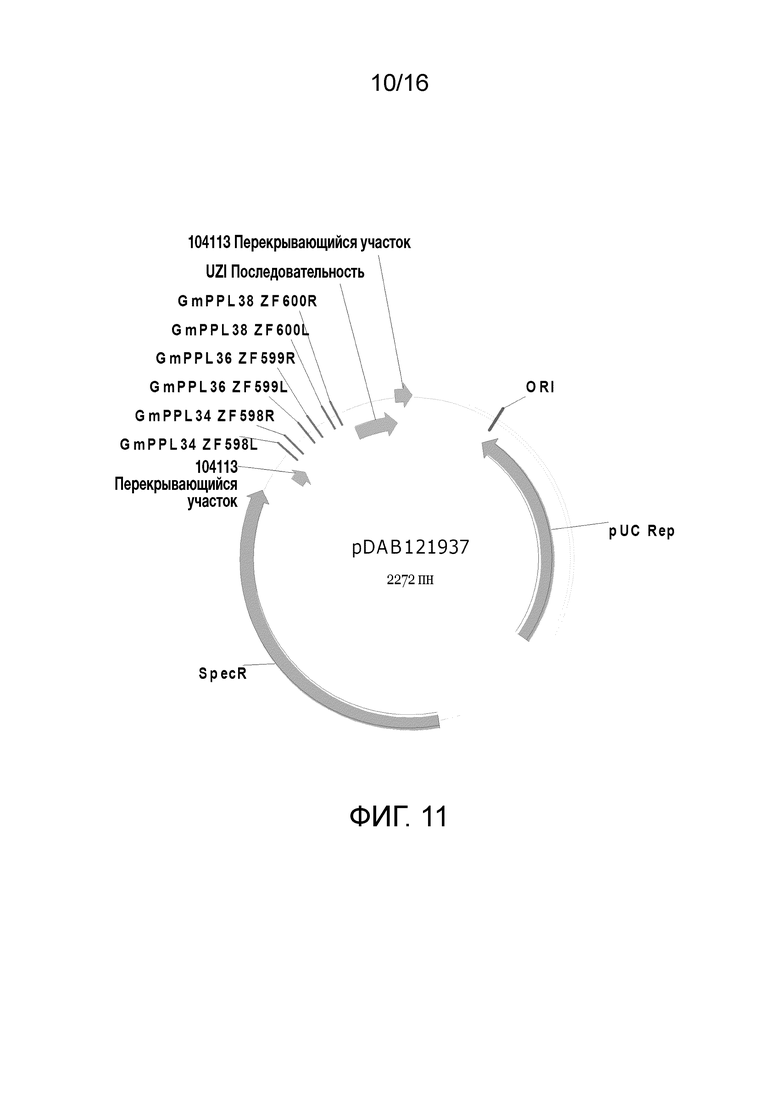
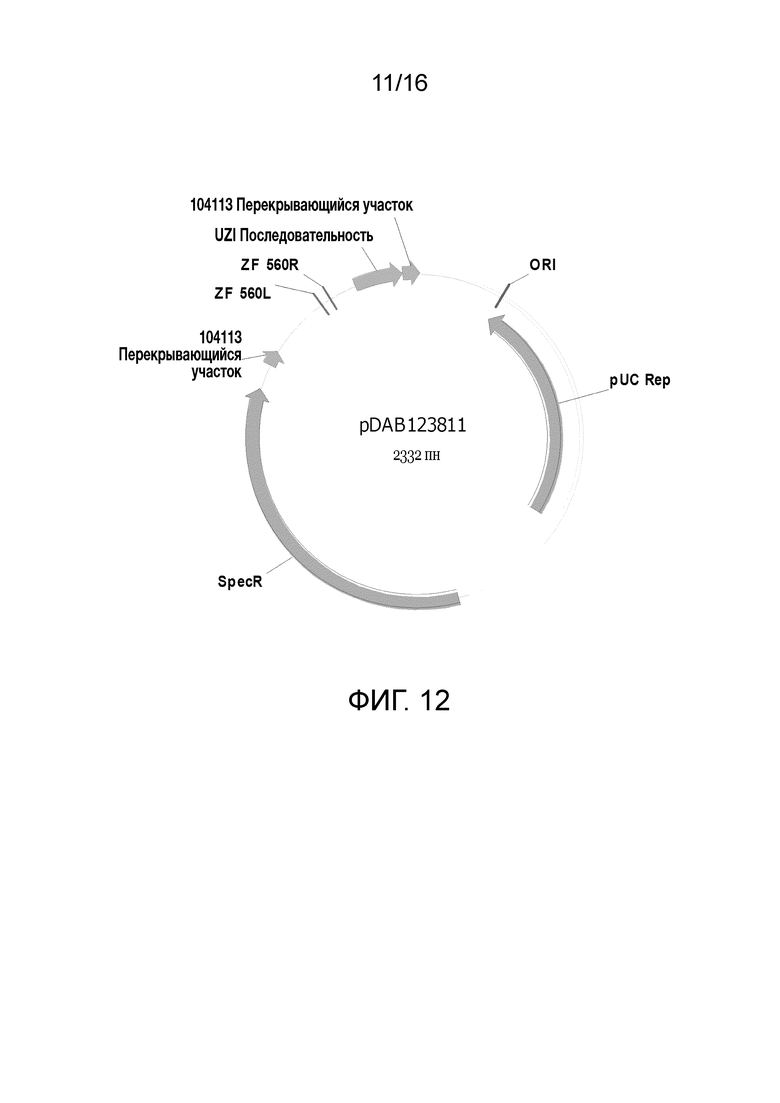
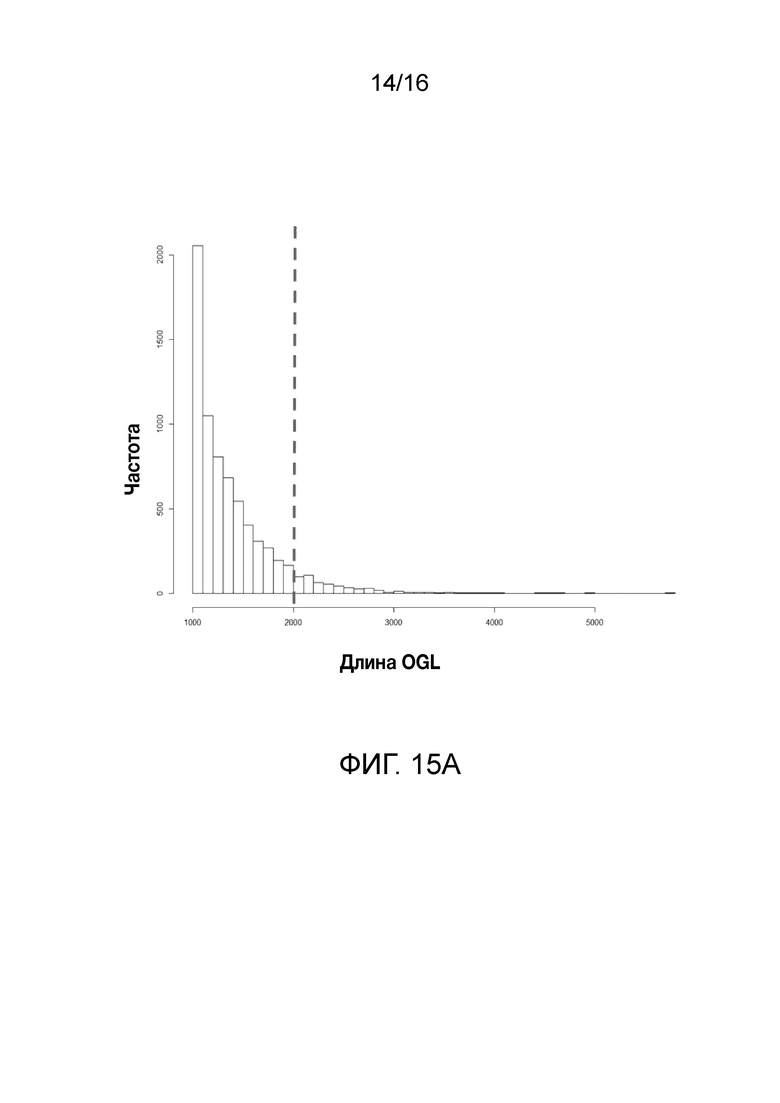
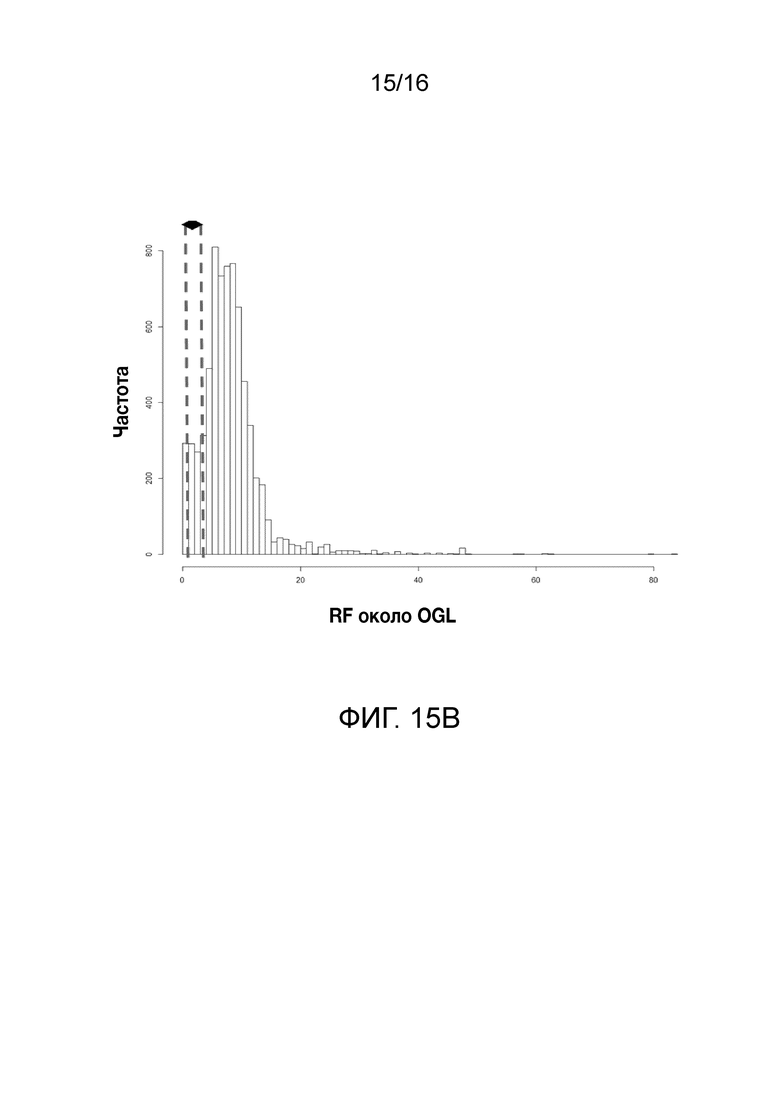
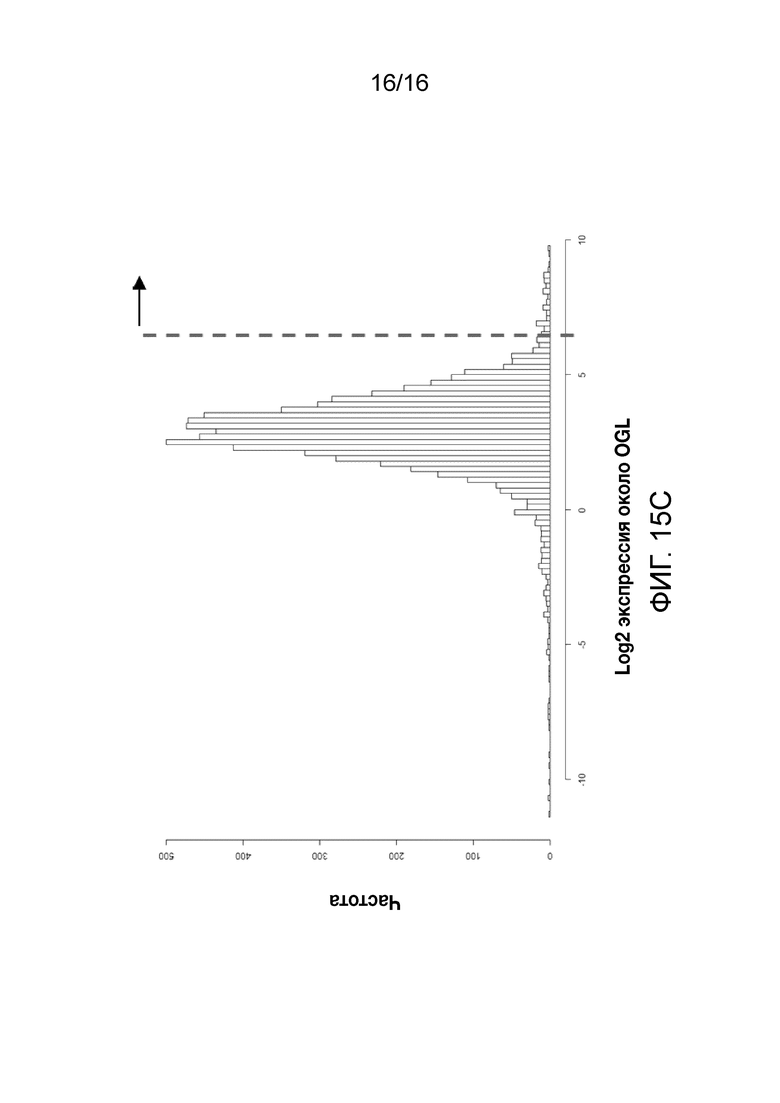
Изобретение относится к области биохимии, в частности к способу получения трансгенной растительной клетки сои. Также раскрыта клетка растения сои. Изобретение позволяет получить трансгенное растение сои, имеющее повышенную экспрессию трансгена. 2 н. и 5 з.п. ф-лы, 15 ил., 12 табл., 6 пр.
1. Способ получения трансгенной растительной клетки сои, включающей целевую ДНК, направленную в одну негенную геномную молекулу нуклеиновой кислоты сои, включающий:
a) отбор негенной геномной молекулы нуклеиновой кислоты сои, где указанная негенная геномная молекула нуклеиновой кислоты имеет длину по меньшей мере 1 тпн и имеет следующие характеристики:
(i) уровень метилирования указанной негенной молекулы нуклеиновой кислоты составляет 1% или меньше;
(ii) указанная негенная молекула нуклеиновой кислоты обладает менее чем 40% идентичностью последовательности с любой другой молекулой нуклеиновой кислоты, содержащейся в геноме сои;
(iii) указанная негенная молекула нуклеиновой кислоты расположена в пределах 40 тпн области известной или предсказанной экспрессируемой кодирующей молекулы нуклеиновой кислоты сои; и
(iv) указанная негенная молекула нуклеиновой кислоты демонстрирует частоту рекомбинации в геноме сои больше 0,01574 сМ/мпн;
b) введение сайт-специфической нуклеазы в растительную клетку, где сайт-специфическая нуклеаза расщепляет указанную негенную геномную молекулу нуклеиновой кислоты сои;
c) введение целевой ДНК в растительную клетку, где указанная целевая ДНК содержит ген, являющийся геном устойчивости к насекомым или геном устойчивости к гербициду;
d) направление целевой ДНК в указанную негенную молекулу нуклеиновой кислоты, где расщепление указанной негенной молекулы нуклеиновой кислоты способствует интеграции целевой ДНК в указанную негенную молекулу нуклеиновой кислоты; и
e) отбор трансгенных растительных клеток, включающих целевую ДНК, введенную в указанную негенную молекулу нуклеиновой кислоты, где указанный способ приводит к получению трансгенной клетки растения сои, имеющей повышенную экспрессию трансгена для вставленного гена устойчивости к насекомым или гена устойчивости к гербициду.
2. Способ получения трансгенной растительной клетки по п. 1, где указанная сайт-специфическая нуклеаза выбрана из группы, состоящей из цинк-пальцевой нуклеазы, нуклеазы CRISPR и TALEN.
3. Способ получения трансгенной растительной клетки по п. 1, где указанная целевая ДНК интегрируется в указанную негенную молекулу нуклеиновой кислоты с помощью метода интеграции посредством направленной гомологией репарации.
4. Способ получения трансгенной растительной клетки по п. 1, где указанная целевая ДНК интегрируется в указанную негенную молекулу нуклеиновой кислоты с помощью метода интеграции посредством негомологичного соединения концов.
5. Способ получения трансгенной растительной клетки по п. 1, где указанная выбранная негенная молекула нуклеиновой кислоты включает следующие характеристики:
a) указанная негенная молекула нуклеиновой кислоты имеет меньше 1% метилирования ДНК в молекуле нуклеиновой кислоты;
b) указанная негенная молекула нуклеиновой кислоты демонстрирует частоту рекомбинации от 0,001574 до 83,52 сМ/мпн в геноме сои;
c) указанная негенная молекула нуклеиновой кислоты демонстрирует уровень занятости нуклеосомами генома сои от 0 до 0,494;
d) указанная негенная молекула нуклеиновой кислоты обладает менее чем 40% идентичностью последовательности с любой другой 1 тпн молекулой нуклеиновой кислоты, содержащейся в геноме сои;
e) указанная негенная молекула нуклеиновой кислоты имеет относительное значение местоположения от 0 до 0,99682 отношения геномного расстояния от центромеры хромосомы сои; и
f) указанная негенная молекула нуклеиновой кислоты имеет процентное содержание гуанина/цитозина от 14,36 до 45,9%.
6. Способ получения трансгенной растительной клетки по п. 5, где известная или предсказанная кодирующая молекула нуклеиновой кислоты сои или молекула нуклеиновой кислоты, включающая 2 тпн область до и 1 тпн область после известного гена, расположена в пределах 40 тпн от указанной негенной молекулы нуклеиновой кислоты.
7. Клетка растения сои, имеющая повышенную экспрессию трансгена для вставленного гена устойчивости к насекомым или гена устойчивости к гербициду, где указанная клетка содержит целевую ДНК, вставленную в негенную молекулу нуклеиновой кислоты, где указанная целевая ДНК содержит ген, кодирующий ген устойчивости к насекомым или ген устойчивости к гербициду, и указанная негенная геномная молекула нуклеиновой кислоты сои имеет длину по меньшей мере 1 тпн и имеет следующие характеристики:
(i) уровень метилирования указанной негенной молекулы нуклеиновой кислоты составляет 1% или меньше;
(ii) указанная негенная молекула нуклеиновой кислоты обладает менее чем 40% идентичностью последовательности с любой другой молекулой нуклеиновой кислоты, содержащейся в геноме сои;
(iii) указанная негенная молекула нуклеиновой кислоты расположена в пределах 40 тпн области известной или предсказанной экспрессируемой кодирующей молекулы нуклеиновой кислоты сои; и
(iv) указанная негенная молекула нуклеиновой кислоты демонстрирует частоту рекомбинации в геноме сои больше 0,01574 сМ/мпн.
| DUFOURMANTEL N | |||
| ET AL.: "Generation of fertile transplastomic soybean", PLANT MOLECULAR BIOLOGY, vol | |||
| Устройство двукратного усилителя с катодными лампами | 1920 |
|
SU55A1 |
| Очаг для массовой варки пищи, выпечки хлеба и кипячения воды | 1921 |
|
SU4A1 |
| Двухколесный автомобиль для формовки кирпичей из разлитой по полю сушки торфяной массы | 1923 |
|
SU478A1 |
| Запор для дверей крытых товарных вагонов | 1923 |
|
SU479A1 |
| YADAV R | |||
| K | |||
| ET AL.: "Enhanced viral intergenic region-specific short interfering RNA accumulation and DNA methylation correlates with resistance against a geminivirus", MOLECULAR PLANT-MICROBE | |||
Авторы
Даты
2020-04-17—Публикация
2014-11-03—Подача