ПЕРЕКРЕСТНЫЕ ССЫЛКИ НА РОДСТВЕННЫЕ ЗАЯВКИ
[0001] Настоящая заявка испрашивает приоритет согласно предварительной заявке на патент США №61/836609, поданной 18 июня 2013 г., и заявке на патент США №61/928390, поданной 16 января 2014 г., в соответствии с § 119(e) раздела 35 Свода законов США (35 U.S.С); обе заявки полностью включены в настоящий документ посредством ссылки.
ССЫЛКА НА ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
[0002] Поданный в электронном формате через систему EFS-Web в соответствии с § 1.821 раздела 37 Свода федеральных правил (37 C.F.R.) одновременно с настоящим документом машиночитаемый (CRF) «Перечень последовательностей», с названием файла 200206_414WO_SEQUENCE_LISTING.txt, включен в настоящий документ посредством ссылки. Электронная версия перечня последовательностей была создана 17 июня 2014 г.; занимаемый на диске объем составляет 222 Кб.
УРОВЕНЬ ТЕХНИКИ
[0003] Молочная кислота, которая может быть получена химическим или биологическим путем, представляет собой химическое соединение, широко используемое в промышленности, от косметической до пищевой, а также фармацевтической и химической промышленности. Поскольку молочная кислота содержит две реакционноспособные функциональные группы, карбоксильную группу и гидроксильную группу, она может подвергаться различным химическим преобразованиям с получением потенциально полезных химических веществ, таких как оксид пропилена, ацетальдегид, акриловая кислота, пропановая кислота, 2,3-пентандион и лактид (Varadarajan and Miller, Biotechnol. Progr. 15:845, 1999). В последнее время повышенное внимание было уделялось применению молочной кислоты для производства полимолочной кислоты (ПМА), которая представляет собой возобновляемый сырьевой материал, используемый при производстве биопластика, представляющий собой более экологически рациональную альтернативу нефтехимическим ресурсам. Оптически чистая молочная кислота может быть полимеризована в высокомолекулярную ПМА путем последовательных реакций поликонденсации, деполимеризации и полимеризации с раскрытием кольца ( and Stolt, Prog. Polym. Sci. 27: 1123, 2002). Полученный полимер ПМА находит применение в разнообразных областях, в том числе для производства защитной одежды, упаковки для пищевых продуктов, мульчирующей пленки, мешков для мусора, жестких контейнеров, термоусадочной пленки и подложек для продуктов с коротким сроком хранения (Drumright et al., Adv. Mater. 12: 1841, 2000; Vink et al., Polym. Degrad. Stabil. 80:403, 2003).
and Stolt, Prog. Polym. Sci. 27: 1123, 2002). Полученный полимер ПМА находит применение в разнообразных областях, в том числе для производства защитной одежды, упаковки для пищевых продуктов, мульчирующей пленки, мешков для мусора, жестких контейнеров, термоусадочной пленки и подложек для продуктов с коротким сроком хранения (Drumright et al., Adv. Mater. 12: 1841, 2000; Vink et al., Polym. Degrad. Stabil. 80:403, 2003).
[0004] Углеводное сырье, используемое в настоящее время для биологического получения лактата, является относительно дорогостоящим. Другие варианты исходного сырья, например, метан, широко доступны и дешевы. Преобразование метана потенциально представляет собой способ значительного снижения стоимости производства молочной кислоты. Однако практический способ достижения указанной цели еще не разработан.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
[0005] Согласно одному варианту реализации в настоящем изобретении предложены не встречающиеся в природе C1-метаболизирующие бактерии, содержащие экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу (ЛДГ).
[0006] Согласно другому варианту реализации в настоящем изобретении предложен не встречающийся в природе С1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу, причем указанный C1-метаболизирующий микроорганизм представляет собой метанотрофный микроорганизм.
[0007] Согласно дополнительному варианту реализации в настоящем изобретении предложен не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу, причем указанный C1-метаболизирующий микроорганизм способен преобразовывать углеродное сырье в лактат, при этом указанное углеродное сырье выбрано из группы, состоящей из метана, диоксида углерода, монооксида углерода, природного газа и сингаза.
[0008] Согласно определенным вариантам реализации в настоящем изобретении предложен не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу, причем указанный C1-метаболизирующий микроорганизм не является дрожжевым.
[0009] Согласно другим вариантам реализации в настоящем изобретении предложен не встречающийся в природе С1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу, причем указанный не встречающийся в природе C1-метаболизирующий микроорганизм способен продуцировать большее количество лактата по сравнению с соответствующим эталонным C1-метаболизирующим микроорганизмом при культивировании в присутствии C1-субстрата по меньшей мере при одной совокупности условий культивирования.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
[0010] На фиг. 1 показан уровень получения лактата для определенных рекомбинантных Methylococcus capsulatus Bath, экспрессирующих гетерологичную нуклеиновую кислоту лактатдегидрогеназы (ldh), по оценке со сравнением OD600 через 72 ч культивирования.
[0011] На фиг. 2 показано соотношение δ13С для разных источников углерода.
[0012] На фиг. 3 показан вектор pMS3 согласно настоящему изобретению, содержащий последовательности, кодирующие белок инициации репликации (trfA) и промотор (Pbla), точку начала репликации (oriV), точку начала переноса (oriT), сайты множественного клонирования (MCS), ген устойчивости к канамицину (KanR) и его промотор (промотор KanR), и точку начала репликации для Е. coli (pUC_Ori_rc).
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
[0013] В настоящем изобретении предложены не встречающиеся в природе C1-метаболизирующие микроорганизмы, и связанные с ними композиции и способы биосинтеза лактата из углеродного сырья. Как правило, указанное углеродное сырье содержит С1-субстрат. Согласно конкретному варианту реализации в настоящем изобретении предложены не встречающиеся в природе C1-метаболизирующие бактерии, содержащие экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу (ЛДГ). В настоящем изобретении также предложен не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу, причем указанный C1-метаболизирующий микроорганизм представляет собой метанотрофный микроорганизм. Согласно конкретному варианту реализации в настоящем изобретении предложен не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу, причем указанный C1-метаболизирующий микроорганизм способен преобразовывать углеродное сырье в лактат. Как правило, указанное углеродное сырье выбрано из группы, состоящей из метана, диоксида углерода, монооксида углерода, сингаза и природного газа. В настоящем изобретении также предложен не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу, причем указанный C1-метаболизирующий микроорганизм не является дрожжевым. Согласно еще одному дополнительному варианту реализации в настоящем изобретении предложен не встречающийся в природе С1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу, причем указанный не встречающийся в природе C1-метаболизирующий микроорганизм способен продуцировать большее количество лактата по сравнению с соответствующим эталонным C1-метаболизирующим микроорганизмом при культивировании в присутствии С1-субстрата по меньшей мере при одной совокупности условий культивирования. Согласно дополнительному варианту реализации в настоящем изобретении предложен не встречающийся в природе С1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу, причем указанный не встречающийся в природе C1-метаболизирующий микроорганизм способен продуцировать большее количество лактата по сравнению с соответствующим эталонным C1-метаболизирующим микроорганизмом при культивировании в присутствии С1-субстрата по меньшей мере при одной совокупности условий культивирования.
[0014] Для понимания настоящего изобретения может быть целесообразно привести, перед переходом к его более подробному описанию, определения некоторых используемых в настоящем документе терминов. Дополнительные определения приведены в различных разделах настоящего описания.
[0015] Предполагается, что в настоящем описании указание любого диапазона описывает любое число в пределах указанного диапазона, и описывает любой диапазон, образуемый любым числом в пределах указанного диапазона, включая конечные точки. В настоящем документе термин «приблизительно» означает ±20% от указанного диапазона, значения или структуры, если не указано иное. Термин «состоящий по существу из» ограничивает объем притязания конкретными материалами или этапами, или не оказывающими существенного влияния на основные и новые характеристики заявленного изобретения. Следует понимать, что термины, используемые в настоящем документе в единственном числе, относятся к «одному или большему количеству» перечисленных компонентов. Использование допускающего альтернативу союза «или» следует понимать как означающее одну из альтернатив, обе альтернативы или любую их комбинацию. В настоящем документе термин «включают», «имеют/обладают» и «содержат» не исключает возможности присутствия других элементов, конкретно не исключенных иным образом.
[0016] В настоящем документе в отношении полипептида термины «лактатдегидрогеназа» и «ЛДГ» используются взаимозаменяемо для обозначения полипептида, обладающего лактатдегидрогеназной активностью, т.е. способного катализировать восстановление пирувата до лактата. Согласно некоторым вариантам реализации указанная лактатдегидрогеназа представляет собой L-лактатдегидрогеназу, которая катализирует восстановление пирувата до L-лактата (ЕС 1.1.1.27). Согласно другим вариантам реализации указанные лактатдегидрогеназа представляет собой D-лактатдегидрогеназу, которая катализирует восстановление пирувата до D-лактата (ЕС 1.1.1.28).
[0017] Термин «углеродное сырье» относится в настоящем документе к любому углеродному(ым) соединению(ям), который(ые) способны метаболизировать не встречающиеся в природе C1-метаболизирующие микроорганизмы согласно настоящему изобретению, такому как, например, C1-субстрат.
[0018] В настоящем документе термин «С1-субстрат» относится к любой углерод-содержащей молекуле, не содержащей углерод-углеродной связи. C1-субстраты могут быть обнаружены в природном газе, природном газе нетипичного происхождения, сингазе, и включают такие молекулы, как, например, метан, метанол, формальдегид, муравьиная кислота (формат), монооксид углерода, диоксид углерода, метилированные амины (например, метиламин, диметиламин, триметиламин и т.п.), метилированные тиолы, метилгалогены (например, бромметан, хлорметан, йодметан, дихлорметан и т.п.), цианид и т.п.
[0019] В настоящем документе термин «метан» относится к простейшему алкановому (C1)-соединению химической формулы CH4, который представляет собой бесцветный газ без запаха при комнатных температуре и давлении.
[0020] В настоящем документе термин «природный газ» относится к встречающимся в природе подземным смесям газов, которые содержат метан. Природный газ может добываться из поровых коллекторов обычными методами (например, бурение, заводнение). Хотя природный газ в основном состоит из метана, он может также содержать другие легкие газообразные алканы (например, этан, пропан, бутан, пентан), диоксид углерода, азот, сульфид водорода или т.п., или любую их комбинацию.
[0021] Термин «природный газ нетипичного происхождения» относится в настоящем документе к встречающимся в природе смесям газов, образовавшихся в пластах с низкой проницаемостью, для доступа к которым необходимо использовать нестандартные методы, такие как гидравлический разрыв, горизонтальное бурение или направленное бурение. Примеры отложений природного газа нетипичного происхождения включают низкопроницаемый песчаный коллектор, сформировавшийся в песчанике или карбонатной породе, метан угольных пластов, сформировавшийся в залежах угля и адсорбированный на угольных частицах, сланцевый газ, сформировавшийся в мелкозернистых сланцевых породах и адсорбированный на глинистых частицах или удерживаемый в мелких порах или микротрещинах, гидраты метана, представляющие собой кристаллическую комбинацию природного газа и воды, образующуюся при низкой температуре и высоком давлении в таких местах, как океанское дно и вечная мерзлота. Состав природного газа нетипичного происхождения, как правило, более изменчив, в том числе потенциально содержит более высокие уровни этана, пропана, бутана, СО2 или любой их комбинации по сравнению со стандартным природным газом.
[0022] В настоящем документе термин «синтез-газ» или «сингаз» относится к полученной синтетическим путем смеси газов, содержащей в основном монооксид углерода (СО) и водород (H2). Сингаз может быть получен, например, путем парового риформинга природного газа или жидких углеводородов, или путем газификации угля, биомассы или отходов. Сингаз может также включать метан, СО2, H2S и другие газы в меньших количествах по сравнению с СО и H2.
[0023] В настоящем документе «C1-метаболизирующий микроорганизм» относится к любому микроорганизму, обладающему способностью использовать (т.е. метаболизировать) С1-субстрат в качестве источника энергии и биомассы, и могут использовать или не использовать другие углеродные субстраты (такие как сахара и сложные углеводы) для получения энергии и биомассы. C1-метаболизирующие микроорганизмы включают бактерии (такие как метанотрофы и метилотрофы) и дрожжи. Согласно определенным вариантам реализации С1-метаболизирующий микроорганизм не включает фотосинтезирующие микроорганизмы, такие как водоросли. Согласно определенным вариантам реализации C1-метаболизирующий микроорганизм является «облигатным C1-метаболизирующим микроорганизмом», что означает, что ему требуются С1-субстраты в качестве источника энергии. Согласно дополнительным вариантам реализации C1-метаболизирующий микроорганизм (например, метанотроф) культивируют в присутствии С1-субстрата (т.е. с применением С1-субстрата в качестве источника энергии).
[0024] В настоящем документе термин «метанотроф», «метанотрофная бактерия» или «метанотрофные бактерии» взаимозаменяемо используются для обозначения метилотрофных бактерий, способных утилизировать метан. Например, в качестве источника углерода и энергии для роста может быть использован метан природного газа. В настоящем документе термин «метанотрофные бактерии» включает «облигатные метанотрофные бактерии», способные утилизировать исключительно метан (например, из природного газа) качестве источников углерода и энергии, и «факультативные метанотрофные бактерии», по своей природе способные использовать, помимо метана, полиуглеродные субстраты, такие как ацетат, пируват, сукцинат, малат или этанол, в качестве источника углерода и энергии.
[0025] В настоящем документе термин «метилотроф» или «метилотрофные бактерии» относится к любым бактериям, способным использовать одноуглеродные соединения (т.е. соединения, не содержащие углерод-углеродных связей). Согласно определенным вариантам реализации метилотрофная бактерия может представлять собой метанотроф. Например, «метанотрофные бактерии» относится к любым метилотрофным бактериям, способным использовать метан в качестве первичного источника углерода и энергии. Примеры метанотрофных бактерий включают Methylomonas, Methylobacter, Methylococcus, Methylosinus, Methylocystis, Methylomicrobium или Methanomonas. Согласно некоторым другим вариантам реализации метилотрофная бактерия представляет собой «облигатную метилотрофную бактерию», которая относится к бактериям, ограниченным утилизацией C1-субстратов для получения энергии.
[0026] В настоящем документе термин «хозяин» относится к клетке или микроорганизму (например, метанотрофу), которые могут быть генетически модифицированы экзогенной нуклеиновой кислотой для получения представляющего интерес полипептида (например, лактатдегидрогеназы). Согласно определенным вариантам реализации клетка-хозяин может необязательно уже содержать или быть модифицирована так, чтобы содержать другие генетические модификации, придающие требуемые свойства, связанные или не связанные с экзогенной ЛДГ, кодируемой экзогенной нуклеиновой кислотой (например, удаление пируватдекарбоксилазы). Например, клетка-хозяин может содержать генетические модификации, которые: минимизируют или уменьшают утилизацию продуцируемого лактатного продукта, минимизируют или уменьшают продуцирование ингибиторов роста клетки-хозяина, обеспечивают усиленный рост, обеспечивают толерантность к загрязняющим примесям или конкретным условиям культивирования (например, кислотостойкость, устойчивость к биоцидам), придают способность метаболизировать дополнительные углеродные субстраты или способность к синтезу дополнительных требуемых продуктов или промежуточных продуктов.
[0027] В настоящем документе термин «молекула нуклеиновой кислоты» и «нуклеиновая кислота» используются взаимозаменяемо для обозначения полимерного соединения, состоящего из ковалентно связанных субъединиц нуклеотидов. Синтетическое получение нуклеиновых кислот включает химические и биологические способы воспроизведения нуклеиновых кислот. Нуклеиновые кислоты включают полирибонуклеиновую кислоту (РНК), полидезоксирибонуклеиновую кислоту (ДНК), любая из которых может быть одноцепочечной или двуцепочечной. ДНК включает кДНК, геномную ДНК, синтетическую ДНК, полусинтетическую ДНК или т.п. Термины «нуклеиновая кислота ЛДГ» и «кодирующая ЛДГ нуклеиновая кислота» используются в настоящем документе взаимозаменяемо для обозначения нуклеиновой кислоты которая кодирует полипептид, обладающий ЛДГ-активностью.
[0028] Термины «полипептид» и «белок» используются взаимозаменяемо в настоящем документе для обозначения полимера аминокислот.
[0029] В настоящем документе термин «рекомбинантный» или «неприродный» (или «не встречающийся в природе») относятся взаимозаменяемо к организму, микроорганизму, клетке, нуклеиновым кислотам или вектору, который(ая,ые) включает(ют) по меньшей мере одно генетическое изменение, или был(а,и) модифицирован(а,ы) путем введения экзогенной нуклеиновой кислоты, или относится к клетке, которая была изменена таким образом, что экспрессию эндогенной(ого) нуклеиновой кислоты или гена можно контролировать, причем такие изменения или модификации вводят или индуцируют посредством генной инженерии. Генетические изменения включают, например, модификации, заключающиеся во введении экспрессируемых нуклеиновых кислот, кодирующих белки или ферменты, или добавлениях, удалениях, заменах других нуклеиновых кислот, или ином функциональном разрушении генетического материала клетки, или модификации, достигаемые классическими методами эволюции штаммов или направленной молекулярной эволюции, известных в данной области техники. Такие модификации включают, например, модификации в кодирующих областях (и их функциональных фрагментах) и некодирующих регуляторных областях, при этом указанные модификации изменяют экспрессию гена или оперона.
[0030] В настоящем документе термин «трансформация» и «трансформирование» относится к введению нуклеиновой кислоты (например, экзогенной или гетерологичной нуклеиновой кислоты) в клетку-хозяина. В трансформированной клетке-хозяин экзогенная или гетерологичная нуклеиновая кислота может располагаться экстрахромосомно или быть интегрирована в хромосому. Интеграция в геном клетки-хозяина и самореплицирующиеся векторы обычно приводит к генетически стабильному наследованию трансформированной молекулы нуклеиновой кислоты. Клетки-хозяева, содержащие трансформированные нуклеиновые кислоты, обозначаются взаимозаменяемыми терминами «рекомбинантные», или «не встречающиеся в природе», или «генетически сконструированные», или «трансформированные», или «трансгенные» (например, бактерии).
[0031] В настоящем документе термин «соответствующий эталонный C1-метаболизирующий микроорганизм» относится к соответствующему C1-метаболизирующему микроорганизму без кодирующей ЛДГ экзогенной нуклеиновой кислоты.
[0032] В отношении нуклеиновой кислоты, полипептида, соединения или активности термины «эндогенный» или «нативный» используются взаимозаменяемо для обозначения нуклеиновой кислоты, полипептида, соединения или активности, присутствующей(ого) в клетке-хозяине дикого типа. При использовании в отношении клетки термин «нативный» относится к клетке дикого типа.
[0033] В настоящем документе термины «гетерологичная» или «экзогенная» нуклеиновая кислота, конструкция или последовательность используются взаимозаменяемо в настоящем документе для обозначения молекулы нуклеиновой кислоты или части молекулы нуклеиновой кислоты, которая не является нативной для клетки-хозяина, или молекулы нуклеиновой кислоты или части молекулы нуклеиновой кислоты, нативной для клетки-хозяина, которая была изменена или мутирована, или молекулы нуклеиновой кислоты, экспрессия которой изменена по сравнению с нативными уровнями экспрессии в аналогичных условиях. Кроме того, термины «гетерологичная» и «экзогенная» может относиться к биологической активности, отличающейся или измененной относительно наблюдаемой эндогенной для клетки-хозяина активности, или не является нативной для клетки-хозяина, и при этом кодируемой молекулой нуклеиновой кислоты, введенной в клетку-хозяина.
[0034] «Процент идентичности» двух или большего количества последовательностей нуклеиновых кислот, или двух или большего количества аминокислотных последовательностей представляет собой функцию от числа идентичных положений в указанных последовательностях (т.е. % идентичности = число идентичных положений / общее число положений × 100), с учетом количества пропусков и длины каждого пропуска, который необходимо ввести для оптимизации выравнивания двух или большего количества последовательностей. Две последовательности оптимально выравнены, если при их выравнивании использованы заданные параметры, т.е. заданную матрицу замен аминокислот, штраф за наличие пропуска (также называемый штрафом за открытие пропуска) и штраф за продолжение пропуска, для получения максимального показателя сходства, возможного для указанной пары последовательностей.
[0035] «Оптимальное выравнивание» для определения процента идентичности двух или большего количества последовательностей осуществляется с применением математического алгоритма, такого алгоритм BLAST (например, Altschul et al., J. Mol. Biol. 275: 403, 1990; см., также BLASTN в сети Интернет по адресу: ncbi.nlm.nih.gov/BLAST). Для аминокислотных последовательностей в качестве дефолтной оценочной матрицы замен в алгоритмах выравнивания аминокислотных последовательностей, таких как BLASTP, используют матрицу BLOSUM62 (Henikoff and Henikoff (1992) Proc. Natl. Acad. Sci. USA 89(22): 10916-10919). Штраф за наличие пропуска применяется при введении одного пропуска аминокислоты в одной из выравниваемых последовательностей; штраф за продолжение пропуска применяется для каждого остатка в пропуске. Оптимальное выравнивание аминокислотных последовательностей проводится с применением BLASTP со следующими параметрами выравнивания: оценочная матрица BLOSUM62, штраф за наличие пропуска = 11, и штраф за продолжение пропуска = 1. Для оптимального выравнивания последовательностей нуклеиновых кислот используют BLASTN со следующими параметрами выравнивания: показатель совпадения/показатель несовпадения = 1/-3, и штраф за наличие пропуска = 5, и штраф за продолжение пропуска = 2. Показатель сходства определяется положениями аминокислот или нуклеотидов каждой последовательности, с которых начинается и которыми заканчивается выравнивание (например, окно выравнивания), и необязательно введением пропуска или нескольких пропусков в одну или обе последовательности для достижения максимального возможного показателя сходства.
[0036] «Консервативной заменой» в данной области техники считается замена одной аминокислоты на другую аминокислоту с аналогичными свойствами. Примеры консервативных замен хорошо известны в данной области техники (см., например, источники: WO 97/09433, стр. 10, опубликована 13 марта 1997 г.; Lehninger, Biochemistry, Second Edition; Worth Publishers, Inc. NY:NY (1975), pp. 71-77; Lewin, Genes IV, Oxford University Press, NY and Cell Press, Cambridge, MA (1990), p. 8).
[0037] В настоящем документе «сверхэкспрессируемый» в отношении нуклеиновой кислоты или белка относится к повышению экспрессии или активности указанной нуклеиновой кислоты или белка. Повышенная экспрессия или активность включает экспрессию или активность нуклеиновой кислоты или белка, повышенную относительно уровня в контрольном или эталонном микроорганизме дикого типа (не являющемся генетически сконструированным). Нуклеиновая кислота или белок сверхэкспрессируется, если указанная экспрессия или активность наблюдается у микроорганизма, для которого не является нормальной экспрессия или активность указанной(ого) нуклеиновой кислоты или белка. Нуклеиновая кислота или белок сверхэкспрессируется, если указанная экспрессия или активность длится или присутствует в течение более длительного периода в рекомбинантном микроорганизме по сравнению с контрольным или эталонным микроорганизмом дикого типа.
[0038] «Ингибировать» или «ингибированный» в настоящем документе относится к изменению, уменьшению, понижающей регуляции, устранению или удалению, прямому или непрямому, экспрессии целевого гена или активности целевой молекулы относительно контрольной, эндогенной или эталонной молекулы, при этом указанное изменение, уменьшение, понижающая регуляция или устранение является статистически, биологически, в промышленном отношении или клинически значимым.
[0039] В настоящем документе термин «производное» относится к модификации соединения химическими или биологическими способами (например, с применением фермента или без применения фермента), при этом модифицированное соединение является структурно аналогичным исходному соединению и (фактически или теоретически) может быть получено из указанного исходного соединения. Производное может обладать иными химическими, биологическими или физическими свойствами, чем исходное соединение, например, быть более гидрофильным или обладать измененной реакционной способностью по сравнению с исходным соединением. Дериватизация (т.е. модификация) может включать замену одного или большего количества фрагментов в молекуле (например, изменение функциональной группы). Например, водород может быть заменен на галоген, такой как фтор или хлор, или гидроксильная группа (-ОН) может быть заменена фрагментом карбоновой кислоты (-СООН). Другие примеры дериватизации включают полимеризацию, гликозилирование, алкилирование, ацилирование, ацетилирование, убиквитинилирование, этерификацию и амидирование.
[0040] Термин «производное» также относится ко всем сольватам, например, гидратам или аддуктам (например, аддуктам спиртов), активным метаболитам и солям исходного соединения. Тип соли зависит от природы фрагментов указанного соединения. Например, кислотные группы, такие как группы карбоновой кислоты, могут образовывать соли щелочных металлов или соли щелочно-земельных металлов (например, соли натрия, соли калия, соли магния, соли кальция, а также соли с физиологически переносимыми ионами четвертичного аммония и соль присоединения кислоты с аммонием и физиологически приемлемыми органическими аминами, такими как, например, триэтиламин, этаноламин или трис-(2-гидроксиэтил)амин). Основные группы могут образовывать соль присоединения кислоты, например, с неорганическими кислотами, такими как соляная кислота, серная кислота или фосфорная кислота, или с органическими карбоновыми кислотами или сульфоновыми кислотами, такими как уксусная кислота, лимонная кислота, молочная кислота, бензойная кислота, малеиновая кислота, фумаровая кислота, винная кислота, метансульфоновая кислота или р-толуолсульфоновая кислота. Соединения, которые одновременно содержат основную группу и кислотную группу, например, карбоксильную группу помимо основных атомов азота, могут находиться в форме цвиттерионов. Соли могут быть получены обычными методами, известными специалистам в данной области техники, например, путем сочетания соединения с неорганической или органической кислотой или основанием в растворителе или разбавителе, или из других солей путем катионного обмена или анионного обмена.
[0041] В настоящем документе термин «лактат» относится ко всем формам молочной кислоты, в том числе все производные формы, такие как соль молочной кислоты, ион молочной кислоты, сложный эфир молочной кислоты, сольват молочной кислоты и/или олигомер молочной кислоты или сложный эфир молочной кислоты.
[0042] Термин «последовательность контроля экспрессии» означает последовательность нуклеиновой кислоты, направляющую транскрипцию нуклеиновой кислоты, с которой она функционально связана, такую как, например, промотор или энхансер.
[0043] Термин «функционально связанный» относится в настоящем документе к конфигурации, в которой контрольная последовательность располагается в надлежащем положении относительно экзогенной кодирующей нуклеиновой кислоты ЛДГ, так что указанная контрольная последовательность влияет на экспрессию указанной кодируемой ЛДГ.
[0044] В настоящем документе термин «соответствующая исходная лактатдегидрогеназа» относится к встречающейся в природе лактатдегидрогеназе или другой известной лактатдегидрогеназе, на которой основан вариант аминокислотной последовательности ЛДГ.
C1-метаболизирующие микроорганизмы - клетки-хозяева
[0045] Не встречающиеся в природе С1-метаболизирующие микроорганизмы согласно настоящему изобретению получают путем введения нужной экзогенной кодирующей нуклеиновой кислоты ЛДГ в нужную клетку-хозяина. C1-метаболизирующий микроорганизм для генетической модификации может представлять собой природный штамм, адаптированный штамм (например, путем проведения ферментации для отбора штаммов с пониженной утилизацией лактата, повышенной скоростью роста или увеличенным общим выходом биомассы по сравнению с исходным штаммом) или ранее модифицированный рекомбинантным образом штамм для преобразования алканов или алкенов в лактат, для понижения или минимизации пируватдекарбоксилазной активности, повышения скорости роста, или любой комбинации перечисленного. Согласно некоторым предпочтительным вариантам реализации указанные С1-метабол изирующие микроорганизмы не являются фотосинтезирующими микроорганизмами, такими как водоросли или растения. Часто C1-метаболизирующий микроорганизм согласно настоящему изобретению не является дрожжами, и как правило, не является грибом. Согласно некоторым вариантам реализации C1-метаболизирующий микроорганизм согласно настоящему изобретению не является фотосинтезирующим микроорганизмом или грибом.
[0046] Согласно определенным вариантам реализации С1-метаболизирующий микроорганизм согласно настоящему изобретению представляет собой прокариотический микроорганизм или бактерию, который(ая) представляет собой генетически модифицированную клетку из одного или большего количества родов Methylomonas, Methylobacter, Methylococcus, Methylosinus, Methylocystis, Methylomicrobium, Methanomonas, Methylophilus, Methylobacillus, Methylobacterium, Hyphomicrobium, Xanthobacter, Bacillus, Paracoccus, Nocardia, Arthrobacter, Rhodopseudomonas или Pseudomonas.
[0047] Согласно дополнительным вариантам реализации указанная C1-метаболизирующая бактерия представляет собой метанотроф или метилотроф. Согласно конкретным вариантам реализации указанный C1-метаболизирующий микроорганизм согласно настоящему изобретению представляет собой метанотроф, который представляет собой генетически модифицированную клетку из одного или большего количества родов Methylomonas, Methylobacter, Methylococcus, Methylosinus, Methylocystis, Methylomicrobium, Methanomonas или Methylocella. Согласно другим вариантам реализации указанный C1-метаболизирующий микроорганизм согласно настоящему изобретению является метилотрофом, который представляет собой генетически модифицированную клетку из одного или большего количества видов Methylobacterium extorquens, Methylobacterium radiotolerans, Methylobacterium populi, Methylobacterium chloromethanicum или Methylobacterium nodulans.
[0048] Согласно некоторым предпочтительным вариантам реализации не встречающийся в природе C1-метаболизирующий микроорганизм согласно настоящему изобретению представляет собой генетически модифицированную метанотрофную бактерию, обладающую способность использовать метан в качестве источника углерода и энергии. Метанотрофные бактерии подразделяют на три группы исходя из путей ассимиляции углерода и структуры внутренней мембраны: тип I (гамма-протеобактерии), тип II (альфа-протеобактерии) и тип X (гамма-протеобактерии). Согласно некоторым вариантам реализации не встречающийся в природе C1-метаболизирующий микроорганизм согласно настоящему изобретению представляет собой генетически модифицированный метанотроф I типа. Согласно некоторым конкретным вариантам реализации не встречающийся в природе C1-метаболизирующий микроорганизм согласно настоящему изобретению представляет собой генетически модифицированный метанотроф II типа. Метанотрофы типа I используют рибулозомонофосфатный (RuMP) путь ассимиляции углерода, тогда как типа II метанотрофы используют сериновый путь. Не встречающийся в природе C1-метаболизирующий микроорганизм согласно настоящему изобретению может представлять собой генетически модифицированный метанотроф X типа. Метанотрофы типа X используют RuMP-путь, однако также экспрессируют незначительные количества ферментов серинового пути.
[0049] Не встречающийся в природе С1-метаболизирующий микроорганизм согласно настоящему изобретению может представлять собой либо генетически модифицированный факультативный метанотроф, либо генетически модифицированный облигатный метанотроф. Например, C1-метаболизирующий микроорганизм согласно настоящему изобретению может представлять собой факультативный метанотроф, который представляет собой генетически модифицированную клетку из одного или большего количества родов и/или видов: Methylocella, Methylocystis и Methylocapsa (например, Methylocella silvestris, Methylocella palustris, Methylocella tundrae, Methylocystis daltona, штамм SB2, Methylocystis bryophila и Methylocapsa aurea KYG), Methylobacterium organophilum (ATCC 27886), Methylibium petroleiphilum или их быстрорастущие варианты. Примеры облигатных метанотрофных бактерий включают Methylococcus capsulatus Bath, Methylomonas 16a (ATCC РТА 2402), Methylosinus trichosporium OB3b (NRRL B-11196), Methylomicrobium buryatense 5G (Таксономический идентификационный №: 675511, Syst. Appl. Microbiol. 24(2): 166-76 (July 2001)), Methylosinus sporium (NRRL B-11197), Methylocystis parvus (NRRL B-11198), Methylomonas methanica (NRRL B-11199), Methylomonas albus (NRRL B-11200), Methylobacter capsulatus (NRRL B-11201), Methylomonas flagellata sp AJ-3670 (FERM P-2400), Methylacidiphilum infernorum, Methylacidiphilum fumariolicum, Methyloacida kamchatkensis, Methylomicrobium alcaliphilum или их быстрорастущие варианты.
[0050] Согласно другим дополнительным вариантам реализации не встречающийся в природе C1-метаболизирующий микроорганизм согласно настоящему изобретению представляет собой сингаз-метаболизирующую бактерию, которая представляет собой генетически модифицированную клетку из одного или нескольких родов: Clostridium, Moorella, Pyrococcus, Eubacterium, Desulfobacterium, Carboxydothermus, Acetogenium, Acetobacterium, Acetoanaerobium, Butyribacterium или Peptostreptococcus. Согласно конкретным вариантам реализации указанный C1-метаболизирующий микроорганизм согласно настоящему изобретению представляет собой сингаз-метаболизирующие бактерии из одного или большего количества видов: Clostridium autoethanogenum, Clostridium ljungdahli, Clostridium ragsdalei, Clostridium carboxydivorans, Butyribacterium methylotrophicum, Clostridium woodii, Clostridium neopropanologen, или их комбинацию.
[0051] Согласно некоторым другим вариантам реализации не встречающийся в природе C1-метаболизирующий микроорганизм согласно настоящему изобретению является эукариотическим, например, дрожжевым организмом, который представляет собой генетически модифицированную клетку из одного или большего количества родов: Candida, Yarrowia, Hansenula, Pichia, Torulopsis или Rhodotorula.
Экспрессионные системы и векторы; способы трансформации
[0052] Любой из рекомбинантных C1-метаболизирующих микроорганизмов, описанных в настоящем документе, может быть трансформирован таким образом, чтобы включать по меньшей мере одну экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу, для обеспечения хозяина новой лактатдегидрогеназой или усиленной лактатдегидрогеназной активности, или может быть генетически модифицирован таким образом, чтобы устранять или существенно уменьшать функцию эндогенного гена, с применением любых из множества способов, известных в данной области техники.
[0053] В данной области техники известен ряд экспрессионных систем и экспрессионных векторов, подходящих для экспрессии гетерологичных нуклеиновых кислот в C1-метаболизирующих микроорганизмах. Согласно определенным вариантам реализации молекула экзогенной нуклеиновой кислоты, кодирующая лактатдегидрогеназу, функционально связана с последовательностью контроля экспрессии, такой как, например, промотор.
[0054] Промоторы, подходящие для применения при практической реализации настоящего изобретения, могут быть конститутивными, не полностью блокируемыми или индуцируемыми, и нативными или ненативными для используемой клетки-хозяина. Примеры промоторов включают пируватдекарбоксилазный (ПДК) промотор, дезоксиксилулозофосфатсинтазный промотор, метанолдегидрогеназный промотор (МДГ) (такой как, например, промотор в межгенной области в 5'-направлении от гена mxaF Methylococcus capsulatus Bath (№ доступа MCA0779) или МДГ-промотор М. extorquens (См., Springer et al., FEMS Microbiol. Lett. 160:119 (1998)), гексулозо-6-фосфатсинтазный промотор, промотор рибосомального белка S16, серингидроксиметилтрансферазный промотор, серинглиоксилатаминотрансферазный промотор, фосфоенолпируваткарбоксилазный промотор, промотор Т5, промотор Trc, промотор синтеза полигидроксиалканоатов (ПГА) (Foellner et al., Appl. Microbiol. Biotechnol. 40:284, 1993), пируватдекарбоксилазный промотор (Tokuhiro et al., Appl. Biochem. Biotechnol. 131:195, 2006), промотор Plac lac-оперона (Toyama et al., Microbiol. 143:595, 1997), гибридный промотор, такой как Ptrc (Brosius et al., Gene 27:161, 1984), промоторы, идентифицированные в природных плазмидах в метилотрофах (ЕР 296484), метанотрофах и т.п.
[0055] Кроме того, могут применяться подходящие гомологичные или гетерологичные промоторы для высоких уровней экспрессии молекул экзогенных нуклеиновых кислот. Например, в патенте США №7098005 описано применение промоторов для высоких уровней экспрессии в присутствии метана или метанола гетерологичной кодирующей нуклеиновой кислоты у C1-метаболизирующих бактерий.
[0056] Согласно определенным вариантам реализации может требоваться регулируемая экспрессия экзогенных нуклеиновых кислот, кодирующих один или большее количество ферментов биосинтеза лактата, для оптимизации скорости роста не встречающихся в природе метанотрофных бактерий и возможности улучшения роста бактерий в условиях различных источников углерода. Это может быть достигнуто путем применения системы с индуцируемым промотором.
[0057] Согласно определенным вариантам реализации нуклеиновая кислота, кодирующая ЛДГ, функционально связана с индуцируемым промотором. Системы с индуцируемым промотором, используемые при практической реализации настоящего изобретения, включают известные в данной области техники, и включают систему с индуцируемым тетрациклином промотором; система с индуцируемым IPTG промотором/промотором lac-оперона, система с индуцируемым тепловым шоком промотором; металлочувствительные промоторные системы; система с индуцируемым нитратами промотором; система с индуцируемым светом промотором; система с индуцируемым экдизоном промотором, индуцируемая/регулируемая система, применение которой описано для метилотрофных и метанотрофных бактерий (см., например, заявку на патент США № US 2010/0221813, включенную в настоящий документ посредством ссылки) и т.п. Например, согласно одному варианту реализации не встречающийся в природе C1-метаболизирующий микроорганизм (например, метанотроф, метилотроф) содержит: (1) экзогенную нуклеиновую кислоту, кодирующую ЛДГ, функционально связанную с промотором, фланкированным последовательностями оператора lacO, и (2) экзогенную нуклеиновую кислоту, кодирующую репрессорный белок lacI, функционально связанную с конститутивным промотором (например, гексулозо-6-фосфатсинтазным промотором). Индуцирование начинается, когда репрессорный белок LacI связывается с последовательностями оператора lacO, фланкирующими промотор ЛДГ или другой промотор, предотвращающий транскрипцию. IPTG связывает репрессор lacI и освобождает его от последовательностей lacO, разрешая транскрипцию. При использовании системы с индуцируемым промотором синтез лактата можно контролировать путем добавления индуктора.
[0058] Экспрессионные системы и экспрессионные векторы, используемые при практической реализации настоящего изобретения, необязательно содержат генетические элементы, такие как, например, один или большее количество сайтов связывания рибосомы для инициации трансляции, сайт терминации транскрипции, сигналы полиаденилирования, сайты рестрикции ферментами, сайты множественного клонирования, другие кодирующие сегменты и т.п.
[0059] Экспрессионные системы и векторы, используемые при практической реализации настоящего изобретения, могут также содержать генетические элементы, облегчающие интеграцию за счет гомологичной либо негомологичной рекомбинации. Генетические элементы облегчающие интеграцию за счет гомологичной рекомбинации, имеют последовательности, гомологичные целевым сайтам интеграции в геномной последовательности нужной клетки-хозяина. Генетические элементы или техники, облегчающие интеграцию за счет негомологичной рекомбинации, включают опосредованную рестриктазными ферментами интеграцию (REMI) (см. источник: Manivasakam et al., Mol. Cell Biol. (1998) 18(3): 1736-1745, включенный в настоящий документ посредством ссылки), опосредованную транспозонами интеграцию и другие элементы и способы, хорошо известные в данной области техники.
[0060] Рекомбинантные способы экспрессии экзогенных или гетерологичных нуклеиновых кислот в микроорганизмах хорошо известны в данной области техники. Описание таких способов можно найти, например, в источниках: Sambrook et al., Molecular Cloning: A Laboratory Manual, Third Ed., Cold Spring Harbor Laboratory, New York (2001); и Ausubel et al., Current Protocols in Molecular Biology, John Wiley and Sons, Baltimore, MD (1999), которые включены в настоящий документ посредством ссылки.
[0061] Согласно определенным вариантам реализации интенсивность и временные характеристики экспрессии лактатдегидрогеназы могут модулироваться с применением способов, известных в данной области техники, для улучшения продуцирования лактата. Например, варьирующая сила промотора или число копий нуклеиновой кислоты может применяться для модуляции уровней экспрессии. В другом примере могут модулироваться временные характеристики экспрессии с применением системы с индуцируемым промотором или полицистронных оперонов. Например, экспрессия ЛДГ может происходить в фазе роста и в стационарной фазе культуры, или только во время стационарной фазы. В другом примере может происходить упорядоченная коэкспрессия ЛДГ и других представляющих интерес генов.
[0062] Введение экзогенной кодирующей ЛДГ нуклеиновой кислоты в клетку-хозяина может осуществляться различными способами, известными в данной области техники. Например, электропорация С1-метаболизирующих бактерий ранее была описана, например, у Toyama et al., FEMS Microbiol. Lett. 166:1, 1998; Kim and Wood, Appl. Microbiol. Biotechnol. 48:105, 1997; Yoshida et al., Biotechnol. Lett. 23:787, 2001, и в опубликованной заявке на патент США №2008/0026005.
[0063] Конъюгация бактерий, которая относится к конкретному типу трансформации, включающей непосредственный контакт донорной и реципиентной клеток, чаще используется для переноса нуклеиновых кислот в С1-метаболизирующие микроорганизмы. Конъюгация бактерий включает смешивание «донорных» и «реципиентных» клеток с обеспечением тесного взаимного контакта. При конъюгация происходит формирование цитоплазматических мостиков между донорными и реципиентными бактериями, с однонаправленной передачей вновь синтезированных донорных молекул нуклеиновых кислот в клетки-реципиенты. Реципиент в реакции конъюгации представлен любой клеткой, способной принимать нуклеиновые кислоты путем горизонтального переноса из донорной бактерии. Донор в процессе конъюгации представляет собой бактерию, которая содержит конъюгативную плазмиду, конъюгативный транспозон или мобилизованную плазмиду. Физический перенос донорной плазмиды может происходить посредством самопередающейся плазмиды или с помощью «хелперной» плазмиды. Конъюгация, задействующая C1-метаболизирующие бактерии, ранее была описана в источниках: Stolyar et al., Mikrobiologiya 64:686, 1995; Motoyama et al., Appl. Micro. Biotech. 42:61, 1994; Lloyd et al, Arch. Microbiol. 171:364, 1999; PCT-публикация № WO 02/18617; и Ali et al., Microbiol. 152:2931, 2006.
[0064] Экспрессия гетерологичных нуклеиновых кислот в C1-метаболизирующих бактерий известна в данной области техники (см., например, патент США №6818424, опубликованную заявку на патент США №2003/0003528). Была описана трансформация метилотрофной бактерии на основе транспозона Mu (Akhverdyan et al., Appl. Microbiol Biotechnol. 91:857, 2011). Была описана система транспозона мини-Tn7 для малокопийной и многокопийной экспрессии гетерологичных нуклеиновых кислот без инсерционной инактивации генов хозяина в Methylobacterium (опубликованная заявка на патент США №2008/0026005).
[0065] Могут требоваться дополнительные генетические модификации C1-метаболизирующего микроорганизма согласно описанию в настоящем документе; они могут осуществляться с применением известных способов. Например, могут применяться различные способы инактивации, нокаутирования или удаления функции эндогенного гена у C1-метаболизирующих бактерий. Аллельный обмен с применением суицидных векторов для конструирования делеционных / инсерционных мутантов у медленно растущих C1-метаболизирующих бактерий также был описан, например, в источниках:Toyama and Lidstrom, Microbiol. 144:183, 1998; Stolyar et al., Microbiol. 145:1235, 1999; Ali et al., Microbiol. 152:2931, 2006; Van Dien et al., Microbiol. 149:601, 2003.
C1-метаболизирующие микроорганизмы - рекомбинантные
[0066] Как описано выше в настоящем документе, C1-метаболизирующие микроорганизмы согласно настоящему изобретению рекомбинантным образом модифицированы так, чтобы включать экзогенные нуклеиновые кислоты, которые экспрессируют или сверхэкспрессируют представляющую(ие) интерес лактатдегидрогеназу(ы), что приводит к получению рекомбинантных микроорганизмов, подходящих для продуцирования лактат. Согласно определенным вариантам реализации в настоящем изобретении предложены не встречающиеся в природе метанотрофные бактерии, содержащие экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу (ЛДГ), при этом указанные метанотрофные бактерии способны преобразовывать углеродное сырье в лактат. Как правило, указанное углеродное сырье представляет собой метан.
[0067] Экзогенные нуклеиновые кислоты используемые при практической реализации настоящего изобретения, могут кодировать встречающуюся в природе или другую известную лактатдегидрогеназу, или вариант последовательности, или усеченный вариант такой соответствующей исходной лактатдегидрогеназы. Такие кодируемые варианты лактатдегидрогеназы могут проявлять повышенную растворимость, экспрессию, стабильность, каталитическую активность, скорость обмена или любую комбинацию перечисленного, или могут представлять собой консервативно модифицированные известные последовательности лактатдегидрогеназы, и последовательности лактатдегидрогеназы, описанные в настоящем документе.
[0068] Кодирующие лактатдегидрогеназу экзогенные нуклеиновые кислоты, подходящие для применения при практической реализации настоящего изобретения, включают последовательности нуклеиновых кислот дикого типа, кодирующие ЛДГ, а также их варианты. Термин «вариант нуклеиновой кислоты» относится в настоящем документе к нуклеиновой кислоте, которая может содержать одну или большее количество замен, добавлений, удалений, вставок, или может представлять собой или содержать фрагмент(ы) эталонной нуклеиновой кислоты. Эталонная нуклеиновая кислота относится к выбранной (исходной нуклеиновой кислоте) дикого типа, кодирующей конкретный фермент ЛДГ (например, LdhA). В силу избыточности генетического кода варианты нуклеиновой кислоты могут влиять или могут не влиять на аминокислотную последовательность.
[0069] Как правило, экзогенные кодирующие ЛДГ нуклеиновые кислоты для введения хозяину согласно описанию в настоящем документе подвергают кодон-оптимизации до введения хозяину для обеспечения эффективной или усиленной экспрессии белка. Оптимизация кодонов относится к изменению кодонов в нуклеиновых кислотах или кодирующих областях нуклеиновых кислот до трансформации для отражения обычной частоты использования кодона у хозяина без изменения полипептида, кодируемой неприродной молекулой ДНК. Способы оптимизации кодонов для оптимальной экспрессии нуклеиновых кислот у гетерологичных хозяев были описаны ранее (см., например, Welch et al., PLoS One 4:e7002, 2009; Gustafsson et al., Trends Biotechnol. 22:346, 2004; Wu et al., Nucl. Acids Res. 35: D76, 2007; Villalobos et al., BMC Bioinformatics 7:285, 2006; опубликованные заявки на патент США №2011/0111413 и №2008/0292918; содержание которых полностью включено в настоящий документ посредством ссылки).
[0070] Вариант нуклеиновой кислоты может кодировать аминокислотную последовательность ЛДГ, содержащую одну или большее количество консервативных замен по сравнению с соответствующей исходной ЛДГ аминокислотной последовательностью. Консервативная замена в полипептиде может происходить естественным образом (например, во встречающихся в природе генетических вариантах) или может быть введена при получении полипептида рекомбинантным образом.
[0071] Подходящие экзогенные нуклеиновые кислоты, кодирующие ЛДГ, которая подходит для применения при практической реализации настоящего изобретения, включают нуклеиновые кислоты, кодирующие ЛДГ из других организмов (т.е. лактатдегидрогеназы и/или кодирующие лактатдегидрогеназу нуклеиновые кислоты, не являющиеся нативными для С1-микроорганизма - хозяина). Такая экзогенная нуклеиновая кислота может кодировать любую из ряда аминокислотных последовательностей ЛДГ, известных в данной области техники. Кроме того, соответствующие кодирующие ЛДГ нуклеиновые кислоты, которые были выделены и клонированы из некоторых из указанных организмов, таких как, например, бактерии, растения, дрожжи, грибы и животные, могут подходить для применения при практической реализации настоящего изобретения. С учетом того, что для сотен организмов известны последовательности полного генома, идентификация нуклеиновых кислот, кодирующих лактатдегидрогеназу у родственных или далеко отстоящих видов, в том числе например, гомологов, ортологов, паралогов и т.п., общеизвестна в данной области техники. Они могут быть кодон-оптимизированы для оптимальной экспрессии у требуемого C1-метаболизирующего микроорганизма с применением известных способов.
[0072] Согласно определенным вариантам реализации указанная молекула экзогенной нуклеиновой кислоты кодирует D- или L-ЛДГ из Actinomyces viscosus, Acinonyx jubatus, Archilochus colubris, Bacillus anthracis, Bacillus caldolyticus, Bacillus coagulans, Bacillus megaterium, Bacillus stearothermophilus (Q9p4b6) (также известного как Geobacillus stearothermophilus), Bacillus subtilis, Bacillus thuringiensis, Bacteroides pectinophilus, Bifidobacterium longum, Bos taurus, Canis familiaris, Canis lupus, Deinococcus radiodurans, Enterococcus faecalis, Enterococcus faecium, Equus ferus, Felis catus, Kluyveromyces lactis, Kluyveromyces maxxianus, Lactobacillus acidophilus, Lactobacillus bulgaricus, Lactobacillus casei, Lactobacillus coryniformis sp. torquens, Lactobacillus delbrueckii (в том числе подвид bulgaricus), Lactobacillus fermentum, Lactobacillus helveticus, Lactobacillus johnsonii, Lactobacillus pentosus, Lactobacillus plantarum, Lactobaillus plantaum, Lactobacillus rhamnosus, Lactococcus lactis, Leuconostoc mesenteroides, Listeria monocytogenes, Listeria marthii, Plasmodium falciparum, Plasmodium ovale, Thermus thermophilus, Mus musculus, Oryctolagus cuniculus, Pediococcus acidilactici, Taeniopygia guttata, Rattus norvegicus, Rhizopus oryzae, Staphylococcus aureus, Streptococcus bovis, Streptococcus pasteurianus, Ruminococcus torques, Staphylococcus simiae, Staphylococcus vitulinus, Staphylococcus lentus, Macrococcus caseolyticus, Bacillus thuringiensis, серологический вариант konkukian, шт. 97-27, Bacillus thuringiensis, серологический вариант chinensis CT-43, Bacillus mycoides и т.п.
[0073] Согласно некоторым вариантам реализации указанная экзогенная нуклеиновая кислота кодирует D-лактатдегидрогеназу. Последовательности многих D-лактатдегидрогеназ известны в данной области техники. Иллюстративные используемые при реализации настоящего изобретения экзогенные нуклеиновые кислоты, которые кодируют D-лактатдегидрогеназу, включают кодирующие D-ЛДГ из Lactobacillus delbrueckii, Lactobacillus plantaum, Lactobacillus johnsonii, Leuconostoc mesenteroides (см., например, заявки JP 2002-136263 А и US 2007/0105202 (SEQ ID NO: 2), обе из которых включены в настоящий документ посредством ссылки); и Lactobacillus helveticus (см., например, заявку WO 2003/102201, которая включена в настоящий документ посредством ссылки). Как правило, указанная экзогенная кодирующая нуклеиновая кислота ЛДГ кодон-оптимизирована для оптимальной экспрессии в конкретной используемой клетке-хозяине.
[0074] В некоторых случаях не встречающийся в природе C1-метаболизирующий микроорганизм содержит экзогенную нуклеиновую кислоту, которая кодирует D-ЛДГ, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно 55%, по меньшей мере приблизительно на 60%, по меньшей мере приблизительно на 65%, по меньшей мере приблизительно на 70%, по меньшей мере приблизительно на 75%, по меньшей мере приблизительно на 80%, по меньшей мере приблизительно на 81%, по меньшей мере приблизительно на 82%, по меньшей мере приблизительно на 83%, по меньшей мере приблизительно на 84%, по меньшей мере приблизительно на 85%, по меньшей мере приблизительно на 86%, по меньшей мере приблизительно на 87%, по меньшей мере приблизительно на 88%, по меньшей мере приблизительно на 89%, по меньшей мере приблизительно на 90%, по меньшей мере приблизительно на 91%, по меньшей мере приблизительно на 92%, по меньшей мере приблизительно на 93%, по меньшей мере приблизительно на 94%, по меньшей мере приблизительно на 95%, по меньшей мере приблизительно на 96%, по меньшей мере приблизительно на 97%, по меньшей мере приблизительно на 98% или по меньшей мере приблизительно 99% идентичную эталонной («исходной ЛДГ») аминокислотной последовательности, выбранной из группы, состоящей из последовательностей SEQ ID NO: 64 и 66, или ее усеченный по N- или С-концу вариант, при этом указанная кодируемая ЛДГ обладает активностью D-ЛДГ, или не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий такую экзогенную нуклеиновую кислоту, продуцирует D-лактат (по оценке, например, с применением способа из примера 1 и заменой в указанном способе набора для анализа D-лактата EnzyFluo™ (Кат. № EFDLC-100, BioAssay Systems, Хейворд, Калифорния 94545) на набор для анализа L-лактата EnzyFluo™). Согласно некоторым вариантам реализации указанная кодируемая D-ЛДГ сохраняет по меньшей мере 50% активности D-ЛДГ по сравнению с исходной ЛДГ. Как правило, не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую такую D-ЛДГ, способен продуцировать большее количество лактата по сравнению с соответствующим эталонным C1-метаболизирующим микроорганизмом, при культивировании в присутствии C1-субстрата по меньшей мере при одной совокупности условий культивирования. Согласно некоторым вариантам реализации не встречающийся в природе C1-метаболизирующий микроорганизм содержит экзогенную нуклеиновую кислоту, кодирующую D-ЛДГ, имеющий последовательность, выбранную из группы, состоящей из SEQ ID NO: 65 и 67, или ее усеченный по N- или С-концу вариант. Согласно конкретным вариантам реализации указанная экзогенная нуклеиновая кислота кодирует D-ЛДГ, имеющую аминокислотную последовательность, соответствующую последовательности SEQ ID N: 64 или SEQ ID NO: 66.
[0075] Согласно другим вариантам реализации не встречающийся в природе C1-метаболизирующий микроорганизм содержит экзогенную нуклеиновую кислоту, кодирующую D-ЛДГ, при этом указанная экзогенная нуклеиновая кислота содержит последовательность нуклеиновой кислоты, по меньшей мере на 50%, 55%, 60%, 65%, 70%, 75%, 80%, 81%, 82%, 83%, 84%, 85%,86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичную последовательности нуклеиновой кислоты, выбранной из группы, состоящей из последовательностей SEQ ID NO: 64 и 66, при этом указанная кодируемая ЛДГ обладает активностью D-ЛДГ, или не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий такую экзогенную нуклеиновую кислоту, продуцирует D-лактат (по оценке, например, с применением способа из примера 1 и заменой в указанном способе набора для анализа D-лактата EnzyFluo™ (Кат. № EFDLC-100, BioAssay Systems, Хейворд, Калифорния 94545) на набор для анализа L-лактата EnzyFluo™). Согласно некоторым вариантам реализации указанная кодируемая D-ЛДГ сохраняет по меньшей мере 50% от активности D-ЛДГ по сравнению с исходной ЛДГ. Как правило, не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую такую D-ЛДГ, способен продуцировать большее количество лактата по сравнению с соответствующим эталонным C1-метаболизирующим микроорганизмом, при культивировании в присутствии C1-субстрата по меньшей мере при одной совокупности условий культивирования.
[0076] Согласно определенным вариантам реализации указанная экзогенная нуклеиновая кислота имеет последовательность, соответствующую последовательности SEQ ID NO: 64 или SEQ ID NO: 66. Согласно некоторым из указанных вариантов реализации не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий такую экзогенную нуклеиновую кислоту способен продуцировать большее количество лактата по сравнению с соответствующим эталонным C1-микроорганизмом, при культивировании в присутствии C1-субстрата по меньшей мере при одной совокупности условий культивирования.
[0077] Согласно дополнительным вариантам реализации не встречающийся в природе C1-метаболизирующий микроорганизм содержит экзогенную нуклеиновую кислоту, кодирующую L-ЛДГ. Примеры нуклеиновых кислот L-ЛДГ включают соответствующие номерам доступа AEEL01000014.1 (область 176996-177985) (Streptococcus bovis), NC_010080.1 (область 957682-958608) (Lactobacillus helveticus), BC146210.1 (Bos taurus), AB776697.1 (Pediococcus acidilactici), EF152288.1 (Rhizopus oryzae), NC_013198.1 (область 619708-620646) (Lactobacillus rhamnosus), NC_010610.1 (область 417799-418740) (Lactobacillus fermentum), NC_008054.1 (область 99905-100828) (Lactobacillus delbrueckii), NC_002662.1 (область 1369224-1370201) (Lactococcus lactis) и NC_004668.1 (область 231275-232258) (Enterococcus faecalis). Например, экзогенные нуклеиновые кислоты, используемые при практической реализации настоящего изобретения, могут кодировать любые из следующих примеров полипептидных последовательностей L-ЛДГ: номера доступа EFM27433.1 (Streptococcus bovis), YP_001577351.1 (Lactobacillus helveticus), AAI46211.1 (Bos taurus), BAM76361.1 (Pediococcus acidilactici), ABL84845.1 (Rhizopus oryzae), YP_003170352.1 (Lactobacillus rhamnosus), YP_001843164.1 (Lactobacillus fermentum), YP_618317.1 (Lactobacillus delbrueckii), NP_267487.1 (Lactococcus lactis), NP_814049.1 (Enterococcus faecalis); SEQ ID NOs: 52 (Staphylococcus simiae CCM 7213), 53 (Staphylococcus vitulinus F1028), 54 (Staphylococcus lentus F1142), 55 (Macrococcus caseolyticus JCSC5402), 56 (Bacillus thuringiensis, серологический вариант konkukian, шт. 97-27), 57 (Bacillus thuringiensis, серологический вариант chinensis СТ-43) и 58 (Bacillus mycoides DSM 2048).
[0078] Согласно некоторым вариантам реализации не встречающийся в природе C1-метаболизирующий микроорганизм содержит экзогенную нуклеиновую кислоту, которая кодирует L-ЛДГ, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно на 55%, по меньшей мере приблизительно на 60%, по меньшей мере приблизительно на 65%, по меньшей мере приблизительно на 70%, по меньшей мере приблизительно на 75%, по меньшей мере приблизительно на 80%, по меньшей мере приблизительно на 81%, по меньшей мере приблизительно на 82%, по меньшей мере приблизительно на 83%, по меньшей мере приблизительно на 84%, по меньшей мере приблизительно на 85%, по меньшей мере приблизительно на 86%, по меньшей мере приблизительно на 87%, по меньшей мере приблизительно на 88%, по меньшей мере приблизительно на 89%, по меньшей мере приблизительно на 90%, по меньшей мере приблизительно на 91%, по меньшей мере приблизительно на 92%, по меньшей мере приблизительно на 93%, по меньшей мере приблизительно на 94%, по меньшей мере приблизительно на 95%, по меньшей мере приблизительно на 96%, по меньшей мере приблизительно на 97%, по меньшей мере приблизительно на 98% и по меньшей мере приблизительно на 99% идентичную эталонной аминокислотной последовательности, выбранной из группы, состоящей из последовательностей SEQ ID NO: 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44, 46, 48, 53, 54, 55, 56, 57, 58, 59 и ее усеченного по N- и С-концу варианта, при этом указанная кодируемая ЛДГ обладает активностью L-ЛДГ, или при этом не встречающийся в природе С1-метаболизирующий микроорганизм, содержащий такую экзогенную нуклеиновую кислоту, продуцирует L-лактат (по оценке, например, с применением способа из примера 1). Согласно некоторым вариантам реализации указанная кодируемая L-ЛДГ сохраняет по меньшей мере 50% активности L-ЛДГ по сравнению с исходной ЛДГ. Как правило, не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую такую L-ЛДГ, способен продуцировать большее количество лактата по сравнению с соответствующим эталонным C1-метаболизирующим микроорганизмом, при культивировании в присутствии C1-субстрата по меньшей мере при одной совокупности условий культивирования.
[0079] Согласно некоторым вариантам реализации эталонная аминокислотная последовательность выбрана из группы, состоящей из последовательностей SEQ ID NO: 2, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44 и 48. Согласно определенным вариантам реализации указанная эталонная аминокислотная последовательность выбрана из группы, состоящей из последовательностей SEQ ID NO: 34, 16 и 24. Примеры экзогенных нуклеиновых кислот включают кодирующие L-ЛДГ, которая имеет аминокислотную последовательность, выбранную из группы, состоящей из последовательностей SEQ ID NO: 2 (ЛДГ Streptococcus pasteurianus), 4 (ЛДГ Lactobacillus helveticus), 6 (вариант ЛДГ Bos taurus), 8 (ЛДГ Ruminococcus torques), 10 (ЛДГ Rhizopus oryzae), 12 (ЛДГ Enterococcus faecalis), 14 (ЛДГ Lactobacillus casei), 16 (ЛДГ Bacillus megaterium), 18 (Taeniopygia guttata ЛДГ), 20 (ЛДГ Lactobacillus plantarum), 22 (ЛДГ Lactobacillus acidophilus), 24 (ЛДГ Staphylococcus aureus), 26 (ЛДГ Bacillus caldolyticus), 28 (ЛДГ Actinomyces viscosus), 30 (ЛДГ Bacillus anthracis), 32 (ЛДГ Bacteroides pectinophilus), 34 (ЛДГ Listeria marthii), 36 (ЛДГ Bacillus subtilis), 38 (ЛДГ Enterococcus faecium), 40 (ЛДГ Bacillus thuringiensis), 42 (Geobacillus stearothermophilus ЛДГ), 44 (ЛДГ Deinococcus radiodurans), 46 (вариант ЛДГ Plasmodium ovale), 48 (ЛДГ Thermus thermophilus), 53 (Staphylococcus simiae CCM 7213), 54 (Staphylococcus vitulinus F1028), 55 (Staphylococcus lentus F1142), 56 (Macrococcus caseolyticus JCSC5402), 57 (Bacillus thuringiensis, серологический вариант konkukian, шт. 97-21), 58 (Bacillus thuringiensis, серологический вариант chinensis CT-43), 59 (Bacillus mycoides DSM 2048), и N- и С-конца ее усеченного варианта. Согласно некоторым вариантам реализации указанная экзогенная нуклеиновая кислота кодирует L-ЛДГ, которая имеет аминокислотную последовательность, выбранную из группы, состоящей из последовательностей SEQ ID NO: 2, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44 и 48. Согласно другим вариантам реализации указанная экзогенная нуклеиновая кислота кодирует L-ЛДГ, которая имеет аминокислотную последовательность, выбранную из группы, состоящей из последовательностей SEQ ID NO: 34, 16 и 24.
[0080] Согласно другим вариантам реализации не встречающийся в природе C1-метаболизирующий микроорганизм содержит экзогенную нуклеиновую кислоту, кодирующую L-ЛДГ, отличающуюся тем, что указанная экзогенная нуклеиновая кислота содержит последовательность нуклеиновой кислоты, по меньшей мере на 50%, 55%, 60%, 65%, 70%, 75%, 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичную эталонной последовательности нуклеиновой кислоты, выбранной из группы, состоящей из последовательностей SEQ ID NO: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 29, 31, 33, 35, 37, 39, 41, 43, 45 и 47, при этом указанная кодируемая ЛДГ обладает активностью L-ЛДГ, или при этом не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий такую экзогенную нуклеиновую кислоту, продуцирует L-лактат (по оценке, например, с применением способа из примера 1). Согласно некоторым вариантам реализации указанная кодируемая L-ЛДГ сохраняет по меньшей мере 50% активности L-ЛДГ по сравнению с исходной ЛДГ. Как правило, не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую такую L-ЛДГ, способен продуцировать большее количество лактата по сравнению с соответствующим эталонным C1-метаболизирующим микроорганизмом, при культивировании в присутствии C1-субстрата по меньшей мере при одной совокупности условий культивирования.
[0081] Согласно определенным вариантам реализации эталонная последовательность нуклеиновой кислоты выбрана из группы, состоящей из последовательностей SEQ ID NO: 1, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43 и 47. Согласно другим вариантам реализации указанная экзогенная нуклеиновая кислота кодирует эталонную последовательность нуклеиновой кислоты, выбранную из группы, состоящей из последовательностей SEQ ID NO: 33, 15 и 23. Иллюстративные примеры экзогенных нуклеиновых кислот, кодирующих L-ЛДГ, включают имеющие последовательность нуклеиновой кислоты, выбранную из группы, состоящей из 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 29, 31, 33, 35, 37, 39, 41, 43, 45 и 47. Согласно некоторым вариантам реализации указанная экзогенная нуклеиновая кислота, кодирующая L-ЛДГ, содержит последовательность нуклеиновой кислоты, выбранную из группы, состоящей из 1, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43 и 47. Согласно некоторым конкретным вариантам реализации указанные экзогенная нуклеиновая кислота, кодирующая L-ЛДГ, содержит последовательность нуклеиновой кислоты, выбранную из группы, состоящей из последовательностей SEQ ID NO: 33, 15 и 23.
[0082] Как правило, усеченные варианты кодируемых ЛДГ согласно описанию в настоящем документе кодируют соответствующую эталонную (т.е. исходную последовательность или вариант исходной последовательности) ЛДГ, которая усечена по N-концу приблизительно на 1-6 аминокислотных остатков (или приблизительно на 1-5 аминокислотных остатков, или приблизительно на 1-4 аминокислотных остатка, или приблизительно на 1-3 аминокислотных остатка, или на 1-2 аминокислотных остатка, или усечена по N-концу на 1 аминокислотный остаток), и/или усечена по С-концу на приблизительно 1-30 аминокислотных остатков (или от приблизительно 1-25, приблизительно 20, приблизительно 15, приблизительно 10 или приблизительно 5 аминокислотных остатков).
[0083] Согласно некоторым вариантам реализации не встречающихся в природе C1-метаболизирующих микроорганизмов, описанных в настоящем документе, не встречающийся в природе C1-метаболизирующий микроорганизм способен продуцировать по меньшей мере приблизительно в 1,5 раз больше лактата по сравнению с соответствующим эталонным C1-метаболизирующим микроорганизмом при культивировании в присутствии С1-субстрата (например, метана) по меньшей мере при одной совокупности условий культивирования. Согласно другим вариантам реализации не встречающийся в природе С1-метаболизирующий микроорганизм способен продуцировать по меньшей мере приблизительно в 2 раза, по меньшей мере приблизительно в 3 раза, по меньшей мере приблизительно в 5 раз, по меньшей мере приблизительно в 10 раз, по меньшей мере приблизительно в 50 раз, по меньшей мере приблизительно в 100 раз, по меньшей мере приблизительно в 500 раз, по меньшей мере приблизительно в 1000 раз больше лактата, и до приблизительно 15000-кратно или приблизительно 20000-кратно большего количества лактата по сравнению с соответствующим эталонным C1-метаболизирующим микроорганизмом при культивировании в присутствии С1-субстрата (например, метана) по меньшей мере при одной совокупности условий культивирования.
[0084] Способы получения экзогенных нуклеиновых кислот, которые кодируют варианты ЛДГ, могут разрабатываться с применением способов на основе филогении, описанных в упоминаемых выше источниках (патент США №8005620; Gustafsson; Welch et al.; Villalobos et al.; Minshull et al.). Каждый вариант ЛДГ, полученной с применением указанных способов сохраняет по меньшей мере 50% активности (предпочтительно 100% или большую активность) и имеет полипептидную последовательность по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% идентичную, или на 100% идентичную эталонной или исходной полипептидной последовательности дикого типа. Согласно определенным вариантам реализации указанные кодируемые варианты ЛДГ включают по меньшей мере одну аминокислотную замену (например, 1, 2, 3, 5, 6, 7, 8, 9, 10 или более, или до 20, 25 или 30 замен) в заданном положении относительно эталонного или исходного фермента дикого типа, при условии, что вариант сохраняет лактатдегидрогеназную активность (например, активность L- или D-лактатдегидрогеназы, или продуцирование L- или D-лактата). Примеры анализов для определения D- и L-лактатдегидрогеназной активности описаны в настоящем документе.
[0085] Согласно некоторым вариантам реализации не встречающийся в природе C1-метаболизирующий микроорганизм согласно настоящему изобретению содержит две или большее количество нуклеиновых кислот, каждая из которых кодирует лактатдегидрогеназу, причем указанные последовательности нуклеиновых кислот и/или последовательностей ЛДГ могут быть одинаковыми или разными. Согласно определенным вариантам реализации несколько копий кодирующей ЛДГ нуклеиновой кислоты вводят в клетку-хозяина, причем может вводиться две, три, четыре, пять, шесть, семь, восемь, девять, десять или большее количество копий одной кодирующей ЛДГ или разных кодирующих ЛДГ нуклеиновых кислот. В случае, когда в C1-метаболизирующий микроорганизм-хозяин вводят две или большее количество молекул экзогенной нуклеиновой кислоты, следует понимать, что указанные две или большее количество молекул экзогенной нуклеиновой кислоты могут быть введены в виде одной молекулы нуклеиновой кислоты (например, на единственном векторе), на разных векторах, интегрированы в хромосому хозяина в одном сайте или нескольких сайтах, и каждый из указанных вариантов реализации тем не менее рассматривается как две или большее количество молекул экзогенной нуклеиновой кислоты. Например, в клетку-хозяина может быть введено более одной молекулы гетерологичной или экзогенной нуклеиновой кислоты, например, в виде отдельных молекул нуклеиновой кислоты, в виде полицистронной молекулы нуклеиновой кислоты, в виде одиночной молекулы нуклеиновой кислоты, кодирующей гибридный белок, или любой их комбинации, и тем не менее это будет рассматриваться как более чем одна гетерологичная или экзогенная нуклеиновая кислота.
[0086] Согласно определенным вариантам реализации указанную экзогенную нуклеиновую кислоту вводят в клетку-хозяин путем конъюгации, трансформации, трансфекции, электропорации или т.п., при этом указанная добавленная молекулы может быть интегрирована в геном хозяина или может существовать в форме экстрахромосомного генетического материала (например, в виде плазмиды или другого самореплицирующегося вектора).
[0087] Согласно определенным вариантам реализации не встречающиеся в природе C1-метаболизирующие микроорганизмы согласно настоящему изобретению также содержат дополнительные генетические модификации. Например, для регуляции экспрессии нативного гена или нуклеиновой кислоты отличным от нормальной экспрессии в природе или культуре нативного гена или нуклеиновой кислоты образом может применяться гетерологичная контрольная последовательность (например, промотор, энхансер). Могут быть осуществлены другие генетические модификации, такие как, например, понижающие или ингибирующие эндогенную пируватдекарбоксилазу, метилглиоксальсинтазу, ЛДГ (например, аэробные ЛДГ, D-ЛДГ), гликогенсинтазу, фосфоглюкомутазу или любую их комбинацию для увеличения продуцирования лактата, выхода лактата, или и первого, и второго. Согласно таким вариантам реализации в неприродных C1-метаболизирующих микроорганизмах (например, неприродной метанотрофной бактерии), содержащих экзогенную нуклеиновую кислоту, кодирующую ЛДГ согласно описанию в настоящем документе, ген эндогенной пируватдекарбоксилазы удален или мутирован таким образом, что наблюдается пируватдекарбоксилазная активность от минимальной до недетектируемой. Согласно определенным вариантам реализации в неприродных C1-метаболизирующих микроорганизмах (например, неприродной метанотрофной бактерии), содержащих экзогенную нуклеиновую кислоту, кодирующую ЛДГ согласно описанию в настоящем документе, эндогенная нуклеиновая кислота, кодирующая метилглиоксальсинтазу или другой фермент метилглиоксального обходного пути удалена или мутирована таким образом, что наблюдается активность указанного метилглиоксального обходного пути от минимальной до недетектируемой, для минимизации накопления метилглиоксаля (токсического побочного продукта).
[0088] Согласно определенным вариантам реализации в неприродных C1-метаболизирующих микроорганизмах (например, неприродной метанотрофной бактерии), содержащих экзогенную нуклеиновую кислоту, кодирующую ЛДГ согласно описанию в настоящем документе эндогенная D-ЛДГ удалена или мутирована, чтобы минимизировать или избежать синтеза рацемической смеси D-лактата и L-лактата, при этом указанный неприродный C1-метаболизирующий микроорганизм продуцирует L-лактат. Согласно определенным вариантам реализации неприродные C1-метаболизирующие микроорганизмы (например, неприродная метанотрофная бактерия) трансформированы двумя, тремя, четырьмя, пятью, шестью, семью, восемью, девятью, десятью или более копиями молекулы экзогенной нуклеиновой кислоты, кодирующей ЛДГ. Согласно дополнительным вариантам реализации неприродные C1-метаболизирующие микроорганизмы (например, неприродные метанотрофная бактерия), содержащие молекулу экзогенной нуклеиновой кислоты, кодирующую ЛДГ согласно описанию в настоящем документе, сверхэкспрессируют ЛДГ в 2-кратном, 3-кратном, 4-кратном, 5-кратном, 6-кратном, 7-кратном, 8-кратном или большем количестве по сравнению с нормальным уровнем экспрессии эндогенной ЛДГ.
[0089] Не встречающиеся в природе C1-метаболизирующие микроорганизмы (например, метанотрофы, метилотрофы) согласно настоящему изобретению могут также быть сконструированы так, чтобы содержать варианты путей или ферментов биосинтеза лактата. Вариации при синтезе лактата могут наблюдаться на одном или большем количестве индивидуальных этапов пути или задействовать совершенно иной путь. Согласно определенным вариантам реализации конкретные реакции лактатного пути катализируют варианты лактатных ферментов или альтернативные лактатные ферменты, которые могут быть скомбинированы с ингибированием или нокаутированием пируватдекарбоксилазной активности, алкогольдегидрогеназной активности, одного или большего количества ферментов метилглиоксального обходного пути, D-ЛДГ, гликогенсинтазы, фосфоглюкомутазы или любой их комбинации. Согласно определенным вариантам реализации используют гибридные пути с нуклеиновыми кислотами, происходящими из двух или большего числа источников, для увеличения продуцирования лактата, выхода лактата, или и первого, и второго.
[0090] В данной области техники известны различные способы ингибирования, инактивации, нокаутирования или удаления функции эндогенного гена в метанотрофных бактериях. Например, разрушение целевого гена представляет собой эффективный способ для подавляющей регуляции гена, когда экзогенную нуклеиновую кислоту встраивают в структурный ген для разрушения транскрипции. Генные кассеты, включающие экзогенную вставочную ДНК (например, генетический маркер), фланкированную последовательностью, обладающей высокой степенью гомологии относительно подлежащей разрушению части целевого гена хозяина, вводят в метанотрофную бактерию - хозяина. Экзогенные ДНК разрушает целевой ген хозяина посредством механизмов нативной репликации ДНК. Аллельный обмен для конструирования делеционных/инсерционных мутантов в С1-метаболизирующих микроорганизмах, в том числе метанотрофных бактериях, был описан, например, в источниках: Toyama and Lidstrom, Microbiol. 144:183, 1998; Stolyar et al., Microbiol. 145:1235, 1999; Ali et al., Microbiol. 152:2931, 2006; Van Dien et al., Microbiol. 149:601, 2003; Martin and Murrell, FEMS Microbiol. Lett. 127:243, 2006. Способы культивирования
Способы получения лактата
[0091] В настоящем документе предложены способы получения лактата, включающие культивирование не встречающегося в природе C1-метаболизирующего микроорганизма (например, метанотрофа, метилотрофа), содержащего экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу, в присутствии углеродного сырья в условиях, подходящих для получения лактата. Как правило, углеродное сырье выбрано из группы, состоящей из метана, метанола, сингаза и природного газа. Чаще указанное углеродное сырье выбрано из группы, состоящей из метана и природного газа. Способы культивирования и поддержания метанотрофных бактериальных культур хорошо известны в данной области техники. В способах получения лактата, например, L-молочной кислоты, ее солей и сложных эфиров могут применяться различные варианты реализации не встречающихся в природе метанотрофных бактерий, описанные в настоящем документе.
[0092] Согласно определенным вариантам реализации лактат синтезируется во время конкретной фазы роста клеток (например, лаг-фазы, логарифмической фазы, стационарной фазы или фазы отмирания). Может быть желательным преобразование углерода из исходного сырья в лактат, а не использование для роста и поддержания C1-метаболизирующего микроорганизма. Согласно некоторым вариантам реализации не встречающиеся в природе C1-метаболизирующие микроорганизмы (например, метанотрофы, метилотрофы) согласно описанию в настоящем документе культивируют до плотности клеток от низкой до средней (OD600), а затем инициируют продуцирование лактата. Согласно некоторым вариантам реализации лактат продуцируется в период, когда C1-метаболизирующий микроорганизм (например, метанотрофная бактерия) уже не делится или делится очень медленно. Согласно некоторым вариантам реализации лактат продуцируется исключительно во время стационарной фазы. Согласно некоторым вариантам реализации лактат продуцируется во время логарифмической фазы и стационарной фазы.
[0093] Композиция в ферментере, содержащая лактат, продуцированный не встречающимся в природе C1-метаболизирующим микроорганизмом (например, метанотрофами, метилотрофами), предложенными в настоящем документе, может также содержать другие органические соединения, связанные с биологическими процессами ферментации. Например, биологические побочные продукты ферментации могут включать один или большее количество спиртов, эпоксидов, альдегидов, кетонов, сложных эфиров, или их комбинацию. Согласно определенным вариантам реализации указанная композиция в ферментере может содержать один или большее количество следующих спиртов: метанол, этанол, бутанол или пропанол. Другие соединения, такие как H2O, СО, CO2, СО N2, H2, O2, и неиспользованное исходное углеродное сырье, такое как метан, этан, пропан и бутан, могут также присутствовать в отходящем газе ферментера.
[0094] Условия, достаточные для получения лактата, включают культивирование не встречающегося в природе C1-метаболизирующего микроорганизма при температуре в диапазоне от приблизительно 25° до приблизительно 50°C. Согласно некоторым вариантам реализации температура культивирования находится в диапазоне от приблизительно 37° до приблизительно 50°C, и может находиться в диапазоне от приблизительно 37°C до приблизительно 45°C. Другие условия, достаточные для получения лактата, включают культивирование не встречающегося в природе C1-метаболизирующего микроорганизма при pH в диапазоне от приблизительно 6 до приблизительно 9, и часто в диапазоне от приблизительно 7 до приблизительно 8.
[0095] Согласно определенным вариантам реализации не встречающиеся в природе C1-метаболизирующие микроорганизмы (например, метанотрофы, метилотрофы), предложенные в настоящем документе, продуцируют лактат в количестве от приблизительно 0,001 г/л культуры до приблизительно 500 г/л культуры. Согласно некоторым вариантам реализации количество полученного лактата составляет от приблизительно 1 г/л культуры до приблизительно 100 г/л культуры. Согласно некоторым вариантам реализации количество полученного лактата составляет от приблизительно 0,001 г/л, 0,01 г/л, 0,025 г/л, 0,05 г/л, 0,1 г/л, 0,15 г/л, 0,2 г/л, 0,25 г/л, 0,3 г/л, 0,4 г/л, 0,5 г/л, 0,6 г/л, 0,7 г/л, 0,8 г/л, 0,9 г/л, 1 г/л, 2,5 г/л, 5 г/л, 7,5 г/л, 10 г/л, 12,5 г/л, 15 г/л, 20 г/л, 25 г/л, 30 г/л, 35 г/л, 40 г/л, 45 г/л, 50 г/л, 60 г/л, 70 г/л, 80 г/л, 90 г/л, 100 г/л, 125 г/л, 150 г/л, 175 г/л, 200 г/л, 225 г/л, 250 г/л, 275 г/л, 300 г/л, 325 г/л, 350 г/л, 375 г/л, 400 г/л, 425 г/л, 450 г/л, 475 г/л или 500 г/л.
[0096] Согласно определенным вариантам реализации лактат представляет собой по существу очищенную жидкость. Способы очищения известны в данной области техники, а оценка чистоты может проводиться с помощью таких методов, как колоночная хроматография, ВЭЖХ или анализ ГХ-МС. Согласно определенным вариантам реализации чистота лактата составляет по меньшей мере 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95% или 99% по массе.
[0097] Согласно определенным вариантам реализации по меньшей мере часть газообразной фазы, остающейся после одного или нескольких этапов выделения лактата, возвращается обратно в систему ферментации.
[0098] Для рекомбинантных метанотрофных бактерий согласно описанию в настоящем документе могут применяться разнообразные методики культивирования. Например, для культивирования метанотрофных бактерий могут применяться методики периодического культивирования или непрерывного культивирования. Согласно определенным вариантам реализации культуры выращивают в аппарате для контролируемого культивирования, например, ферментере, биореакторе, половолоконном мембранном биореакторе или т.п.
[0099] Классический способ периодического культивирования представляет собой закрытую систему, состав среды в которой задается в начале культивирования и не претерпевает изменений извне во время процесса культивирования. Соответственно, в начале процесса культивирования среду инокулируют нужным C1-метаболизирующим микроорганизмом (например, метанотрофом), и оставляют для роста или метаболической активности без добавления чего-либо в систему. Однако, как правило, «периодическая» культура является периодической в отношении добавления источника углерода, и часто предпринимаются попытки контролировать такие факторы, как показатель pH и концентрация кислорода. В периодических системах состав метаболитов и биомассы в системе постоянно изменяются вплоть до момента прекращения культивирования. В периодических культурах происходит умеренный рост клеток в статической лаг-фазе до наступления логарифмической фазы активного роста, и, наконец, стационарной фазы, когда рост замедляется или останавливается. Без вмешательства клетки в стационарной фазе в конечном итоге гибнут. Клетки в фазе логарифмического роста часто отвечают за массовую продукцию конечного продукта или промежуточного продукта в некоторых системах. В других системах продуцирование может происходить в стационарной или постэкспоненциальной фазе.
[0100] Подпитываемая система представляет собой вариант стандартной периодической системы. Процессы подпитываемого культивирования включают обычную периодическую систему с модификацией, заключающейся в поэтапном добавлении субстрата по мере развития культуры. Подпитываемые системы подходят для применения в тех случаях, когда наблюдается тенденция к ингибированию метаболизма клеток за счет катаболитной репрессии, и если требуется ограничить количество субстрата в среде. Фактическую концентрацию субстрата в подпитываемых системах измерить сложно, поэтому ее оценивают на основании изменений измеряемых факторов, таких как pH, растворенный кислород и парциальное давление отработанных газов, таких как CO2. Способы периодического и подпитываемого культивирования широко распространены и известны в данной области техники (см., например, источники: Thomas D. Brock, Biotechnology: A Textbook of Industrial Microbiology, 2nd Ed. (1989) Sinauer Associates, Inc., Sunderland, MA; Deshpande, Appl. Biochem. Biotechnol. 36:221, 1992), включенные в настоящий документ посредством ссылки.
[0101] Непрерывные культуры представляют собой «открытые» системы, в которых определенную культуральную среду постоянно добавляют в биореактор, и равное количество кондиционированной среды одновременно извлекают для переработки. В непрерывных культурах обычно поддерживается постоянная высокая плотность клеток в жидкой фазе, при этом клетки находятся в основном в логарифмической фазе роста. Как вариант, может осуществляться непрерывное культивирование иммобилизованных клеток, при котором непрерывно добавляют углерод и питательные вещества, а ценные продукты, побочные продукты и отходы непрерывно удаляют из клеточной массы. Иммобилизация клеток может осуществляться с применением разнообразных твердых подложек, состоящих из природных и/или синтетических материалов.
[0102] Непрерывная или полунепрерывная культура позволяет обеспечить модуляцию одного фактора или любого числа факторов, которые влияют на рост клеток или концентрацию конечного продукта. Например, в одном из способов поддерживается ограниченный фиксированный уровень питательного вещества, например, источника углерода, или уровень азота, а все другие параметры могут варьировать. В других системах ряд факторов, влияющих на рост, может непрерывно изменяться, при поддержании наряду с этим постоянной концентрации клеток, измеряемой по непрозрачности среды. Непрерывные системы направлены на поддержание условий стационарного роста и, соответственно, потеря клеток в результате удаления среды должна компенсироваться скоростью роста клеток в культуре. Способы модуляции питательных веществ и факторов роста в процессах непрерывного культивирования, а также техники максимизации скорости образования продукта, хорошо известны в данной области техники, и различные способы подробно описаны у Brock, выше.
[0103] Жидкофазные биореакторы (например, реактор с перемешиванием, с фильтрующим наполнителем, однофазный жидкостный реактор, двухфазный жидкостный реактор, половолоконный мембранный реактор) хорошо известны в данной области техники и могут применяться для культивирования не встречающихся в природе микроорганизмов и биокатализа.
[0104] При применении газофазных биореакторов, субстраты для биопроизводства поглощаются не встречающимися в природе микроорганизмами, клеточными лизатами или их бесклеточными фракциями из газа, а не из жидкости. Применение газофазных биореакторов для микроорганизмов известно в данной области техники (см., например, источники: патенты США №2793096; №4999302; №5585266; №5079168; и №6143556; удостоверение об изобретении США Н1430; опубликованную заявку на патент США №2003/0032170; Emerging Technologies in Hazardous Waste Management III, 1993, eds. Tedder and Pohland, pp. 411-428, все из которых включены в настоящий документ посредством ссылки). Примеры газофазных биореакторов включают прямоточную систему, насосную систему с замкнутым контуром, и реактор с псевдоожиженным слоем. При применении газофазных биореакторов метан или другие газообразные субстраты могут быть легко использованы для биоконверсии полипептидами, обладающими, например, монооксигеназной активностью. Согласно определенным вариантам реализации способы преобразования газа в лактатную композицию реализуют в газофазных биореакторах. Согласно дополнительным вариантам реализации способы преобразования газа в лактатную композицию реализуют в реакторах с псевдоожиженным слоем. В реакторе с псевдоожиженным слоем текучая среда (т.е. газ или жидкость) поднимется наверх через слой частиц носителя, обычно - песка, гранулированного активированного угля или диатомовой земли, на которых могут прикрепляться и расти микроорганизмы. Скорость текучей среды такова, что слой частиц носителя с присоединенным микроорганизмами суспендирован (т.е. псевдоожижен). Микроорганизмы, присоединенные к слою частиц носителя, свободно циркулируют в текучей среде, обеспечивая эффективную массопередачу субстратов в текучей среде микроорганизмам и усиленный рост микроорганизмов. Примеры реакторов с псевдоожиженным слоем включают поршневые проточные биореакторы и реакторы полного смешения. Применение реакторов с псевдоожиженным слоем с микробными биопленками известно в данной области техники (например, Pfluger et al., Bioresource Technol. 102:9919, 2011; Fennell et al., Biotechnol, Bioengin. 40:1218, 1992; Ruggeri et al., Water Sci. Technol. 29:347, 1994; патенты США №4032407; 4009098; 4009105; и 3846289, все из которых включены в настоящий документ посредством ссылки).
[0105] Метанотрофные бактерии, описанные в настоящем описании, могут культивироваться в виде изолированной чистой культуры, с гетерологичными неметанотрофным(и) микроорганизмом(ами), которые могут благоприятствовать росту, или может быть скомбинирован с одним или большим числом других штаммов или видов метанотрофных бактерий для получения смешанной культуры.
[0106] Согласно альтернативным вариантам реализации в описанных в настоящем документе способах используются рекомбинантные C1-метаболизирующие микроорганизмы согласно настоящему изобретению или их клеточные лизаты, иммобилизованные на твердой основе, внутри твердой основы или за твердой основой. Согласно дополнительным вариантам реализации не встречающиеся в природе О микроорганизмы согласно настоящему изобретению, клеточные лизаты или их бесклеточные экстракты находятся по существу в неводном состоянии (например, лиофилизированы). Рекомбинантные микроорганизмы, клеточные лизаты или их бесклеточные фракции временно или постоянно прикреплены к твердой основе, внутри твердой основы или за твердой основой в биореакторе. Питательные вещества, субстраты и другие необходимые факторы добавляют в твердые основы, таким образом, чтобы клетки могли катализировать требуемые реакции. Рекомбинантные микроорганизмы могут расти на поверхности твердой основы (например, в виде биопленки). Рекомбинантные микроорганизмы, клеточные лизаты или полученные из них бесклеточные фракции могут быть прикреплены к поверхности или внутри твердой основы без клеточного роста или в неживом состоянии. Примеры твердых основ-подложек для микроорганизмов включают полипропиленовые кольца, керамические биокольца, керамические седла, волокнистые подложки (например, мембранные), гранулы из пористого стекла, полимерные гранулы, древесный уголь, активированный уголь, высушенный силикагель, частицы оксида алюминия, оттавский песок, глина, полиуретановые листовые носители для клеток и псевдоожиженный носитель на основе частиц (например, песок, гранулированный активированный уголь, диатомовая земля, гранулы геля альгината кальция).
[0107] Лактат, полученный с применением композиций и способов, описанных в настоящем документе, может быть дополнительно обработан с получением других высокоценных продуктов с применением способов, известных в данной области техники. Например, после выделения или очищения лактат может быть изменен или дериватизирован для различных применений, например, для изготовления биопластиков.
[0108] В настоящем изобретении также предложены подходящие продукты, которые включают лактат и содержащие его композиции, и не встречающиеся в природе C1-метаболизирующие микроорганизмы согласно настоящему изобретению. Композиции согласно настоящему изобретению могут быть представлены в твердой форме (например, в виде порошка) или жидкой форме (например, в виде раствора, эмульсии, суспензии и т.п.) и могут содержать лактат в комбинации с компонентом, выбранным из группы, состоящей из не встречающегося в природе C1-метаболизирующего микроорганизма согласно настоящему изобретению, воды, соли, кислоты, основания, буфера и т.п.
[0109] Лактат и содержащие его композиции, полученные с применением способов, предложенных в настоящем документе, можно отличить от лактата, полученного из нефтехимических веществ, или от лактата, полученного биосинтезом с использованием неметанотрофных бактерий, по изотопной сигнатуре углерода. На существующем уровне техники измерение стабильных изотопов и подходы на основе массового баланса широко используются для оценки глобальных источников и резервуаров метана (см., Whiticar and Faber, Org. Geochem. 10:759, 1986; Whiticar, Org. Geochem. 16:531, 1990). Мера степени изотопного фракционирования углерода, вызванного микробиологическим окислением метана, может быть определена путем измерения показателя изотопного состава (т.е. отношения стабильных изотопов 13С:12С) остаточного метана. Например, аэробные метанотрофы могут метаболизировать метан в помощью специфического фермента метанмонооксигеназы (ММО). Метанотрофы преобразуют метан в метанол и затем в формальдегид. Формальдегид может быть далее окислен до CO2 для обеспечения клетки энергией в форме восстановительных эквивалентов (NADH), или включен в биомассу либо через рибулозомонофосфатный (RuMP), либо через сериновый цикл (Hanson and Hanson, Microbiol. Rev. 60:439, 1996), которые являются прямыми аналогами путей ассимиляции углерода у фотосинтезирующих организмах. В частности, метанотроф I типа использует RuMP-путь для синтеза биомассы и производит биомассу полностью из CH4, тогда как метанотроф II типа использует сериновый путь, ассимилирующий 50-70% углерода клеток из СН4 и 30-50% из СО2 (Hanson and Hanson, 1996). Способы измерения изотопного углеродного состава предложены, например, Templeton et al. (Geochim. Cosmochim. Acta 70:1739, 2006); указанные способы полностью включены в настоящий документ посредством ссылки. Отношение стабильных изотопов углерода 13С/12С в лактате (выраженное в виде значения δ13С в тысячных долях, ‰), варьирует в зависимости от источника и чистоты используемого С1-субстрата (см., например, фиг. 2).
[0110] Например, отношение δ13С в лактате, происходящем из нефти, составляет от приблизительно - 22‰ до приблизительно - 24‰. Отношение δ13С в лактате, биосинтезированном в основном из полученной из кукурузы глюкозы (δ13С - 10,73‰), составляет от приблизительно - 14,66‰ до - 14,85‰. Показатели δ13С в лактате, биосинтезированном из углерода возобновляемых источников, предположительно имеют менее отрицательные значения по сравнению с лактатом, происходящим из нефти. Так или иначе, отношение δ13С в метане природного газа отличается от значений для большинства источников углерода, более негативными значениями отношения δ13С, чем в неочищенной нефти. Метанотрофные бактерии, согласно наблюдениям, используют предпочтительно 12С, и уменьшение поглощения ими 13С в условиях избытка метана приводит к еще более выраженного сдвигу значения δ13С в отрицательную область. Отношение δ13С в лактате, продуцированном метанотрофными бактериями согласно описанию в настоящем документе, принимает более отрицательные значения, чем в лактате из неочищенной нефти или возобновляемых источников углерода, варьируя в диапазоне от приблизительно - 30‰ до приблизительно - 70‰.
[0111] Согласно определенным вариантам реализации отношение δ13С в лактатной композиции (т.е. композиции, содержащей лактат) и содержащемся в ней лактате составляет менее чем приблизительно - 30‰, - 40‰ или - 50‰. Согласно определенным вариантам реализации отношение δ13С в указанной лактатной композиции и содержащемся в ней лактате составляет от приблизительно - 30‰ до приблизительно - 40‰, или от приблизительно - 40‰ до приблизительно - 50‰. Согласно дополнительным вариантам реализации δ13С в указанной лактатной композиции и содержащемся в ней лактате составляет менее чем - 30‰, менее чем - 31‰, менее чем - 32‰, менее чем - 33‰, менее чем - 34‰, менее чем - 35‰, менее чем - 36‰, менее чем - 37‰, менее чем - 38‰, менее чем - 39‰, менее чем - 40‰, менее чем - 41‰, менее чем - 42‰, менее чем - 43‰, менее чем - 44‰, менее чем - 45‰, менее чем - 46‰, менее чем - 47‰, менее чем - 48‰, менее чем - 49‰, менее чем - 50‰, менее чем - 51‰, менее чем - 52‰, менее чем - 53‰, менее чем - 54‰, менее чем - 55‰, менее чем - 56‰, менее чем - 57‰, менее чем - 58‰, менее чем - 59‰, менее чем - 60‰, менее чем - 61‰, менее чем - 62‰, менее чем - 63‰, менее чем - 64‰, менее чем - 65‰, менее чем - 66‰, менее чем - 67‰, менее чем - 68‰, менее чем - 69‰ или менее чем - 70‰. Согласно конкретным вариантам реализации лактат в таких вариантах реализации может быть представлен свободной кислотой (а именно, молочной кислотой, СН3СН(ОН)СООН), причем δ13С в такой свободной кислоте соответствует описанному выше в настоящем документе. Согласно другим вариантам реализации указанный лактат может представлять собой соль, ангидрид, олигомер или сложный эфир, который содержит одну или большее количество СН3С(ОН)С(O)-групп (как в случае соли или сложного эфира), или одну или большее количество СН3С(O-)С(O)-)-групп (как в случае некоторых сложных эфиров, олигомеров и ангидридов), атомы углерода лактатной группы которой(ых) имеют показатель δ13С согласно описанию в настоящем документе.
[0112] Варианты реализации настоящего изобретения включают следующие:
1. Не встречающийся в природе C1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу (ЛДГ), при этом указанный C1-метаболизирующий микроорганизм способен преобразовывать углеродное сырье в лактат.
2. C1-метаболизирующий микроорганизм согласно варианту реализации 1, при этом указанная молекула нуклеиновой кислоты, кодирующая ЛДГ, происходит из Actinomyces viscosus, Acinonyx jubatus, Archilochus colubris, Bacillus anthracis, Bacillus caldolyticus, Bacillus coagulans, Bacillus megaterium, Bacillus stearothermophilus (Q9p4b6) (также известного как Geobacillus stearothermophilus), Bacillus subtilis, Bacillus thuringiensis, Bacteroides pectinophilus, Bifidobacterium longum, Bos taurus, Canis familiaris, Canis lupus, Deinococcus radiodurans, Enterococcus faecalis, Enterococcus faecium, Equus ferus, Felis catus, Kluyveromyces lactis, Kluyveromyces maxxianus, Lactobacillus acidophilus, Lactobacillus bulgaricus, Lactobacillus casei, Lactobacillus coryniformis sp.torquens, Lactobacillus delbrueckii (в том числе подвида bulgaricus), Lactobacillus fermentum, Lactobacillus helveticus, Lactobacillus johnsonii, Lactobacillus pentosus, Lactobacillus plantarum, Lactobacillus rhamnosus, Lactococcus lactis, Listeria monocytogenes, Plasmodium falciparum, Plasmodium ovale, Thermus thermophilus, Mus musculus, Oryctolagus cuniculus, Pediococcus acidilactici, Taeniopygia guttata, Rattus norvegicus, Rhizopus oryzae, Staphylococcus aureus или Streptococcus bovis.
3. C1-метаболизирующий микроорганизм согласно варианту реализации 1, отличающийся тем, что указанная последовательность экзогенной нуклеиновой кислоты, кодирующая ЛДГ, кодон-оптимизирована для экспрессии в метанотрофной бактерии.
4. С1-метаболизирующий микроорганизм согласно варианту реализации 1, при этом указанная молекула экзогенной нуклеиновой кислоты кодирует аминокислотную последовательность, соответствующую представленной в любой из последовательностей SEQ ID NO: 2, 4, 6, 8, 10, 60, 61, 62, 63 или 12.
5. С1-метаболизирующий микроорганизм согласно варианту реализации 1, отличающийся тем, что указанная экзогенная нуклеиновая кислота, кодирующая ЛДГ, имеет последовательность, соответствующую представленной в любой из последовательностей SEQ ID NO: 1, 3, 5, 7, 9 или 11.
6. C1-метаболизирующий микроорганизм согласно варианту реализации 1, отличающийся тем, что указанная экзогенная нуклеиновая кислота, кодирующая ЛДГ, функционально связана с последовательностью контроля экспрессии.
7. C1-метаболизирующий микроорганизм согласно варианту реализации 6, при этом указанный последовательность контроля экспрессии представляет собой промотор, выбранный из пируватдекарбоксилазного промотора, метанолдегидрогеназного промотора, гексулозо-6-фосфатсинтазного промотора, промотора рибосомального белка S16, серингидроксиметилтрансферазного промотора, серинглиоксилатаминотрансферазного промотора, фосфоенолпируваткарбоксилазного промотора, промотора Т5 или промотора Trc.
8. C1-метаболизирующий микроорганизм согласно варианту реализации 1, отличающийся тем, что указанный микроорганизм обладает минимальной, недетектируемой или нокаутированной эндогенной пируватдекарбоксилазной активностью.
9. C1-метаболизирующий микроорганизм согласно варианту реализации 1, отличающийся тем, что указанный микроорганизм содержит один или большее количество эндогенных ферментов метилглиоксалевого обходного пути, который(ые) обладает(ют) минимальной активностью или нокаутирован(ы).
10. С1-метаболизирующий микроорганизм согласно варианту реализации 1, отличающийся тем, что указанный микроорганизм обладает минимальной, недетектируемой или нокаутированной эндогенной алкогольдегидрогеназной активностью.
11. С1-метаболизирующий микроорганизм согласно варианту реализации 1, отличающийся тем, что указанный микроорганизм обладает пониженной или нокаутированной активностью эндогенной пируватдекарбоксилазы, алкогольдегидрогеназы, метилглиоксальсинтазы или любой их комбинации.
12. С1-метаболизирующий микроорганизм согласно варианту реализации 1, отличающийся тем, что указанный микроорганизм способен продуцировать от приблизительно 1 мг/л до приблизительно 500 г/л лактата.
13. С1-метаболизирующий микроорганизм согласно любому из вариантов реализации 1-12, при этом указанный С1-метаболизирующий микроорганизм представляет собой метанотрофный микроорганизм.
14. С1-метаболизирующий микроорганизм согласно варианту реализации 13, при этом указанный метанотроф представляет собой Methylomonas, Methylobacter, Methylococcus, Methylosinus, Methylocystis, Methylomicrobium, Methanomonas, Methylocella или Methylocapsa.
15. С1-метаболизирующий микроорганизм согласно варианту реализации 13, при этом указанный метанотроф представляет собой Methylococcus capsulatus, шт. Bath, Methylomonas methanica 16a (ATCC РТА 2402), Methylosinus trichosporium OB3b (NRRL B-11196), Methylosinus sporium (NRRL B-11197), Methylocystis parvus (NRRL B-11198), Methylomonas methanica (NRRL B-11199), Methylomonas albus (NRRL B-11200), Methylobacter capsulatus (NRRL B-11201), Methylobacterium organophilum (ATCC 27886), Methylomonas sp AJ-3670 (FERM P-2400), Methylocella silvestris, Methylocella palustris (ATCC 700799), Methylocella tundrae, Methylocystis daltona, штамм SB2, Methylocystis bryophila, Methylocapsa aurea KYG, Methylacidiphilum infernorum, Methylacidiphilum fumariolicum, Methyloacida kamchatkensis, Methylibium petroleiphilum или Methylomicrobium alcaliphilum.
16. C1-метаболизирующий микроорганизм согласно любому из вариантов реализации 1-15, отличающийся тем, что углеродное сырье представляет собой метан, метанол, сингаз, природный газ или природный газ нетипичного происхождения.
17. Способ получения лактата, включающий культивирование не встречающегося в природе С1-метаболизирующего микроорганизма, содержащего молекулу экзогенной нуклеиновой кислоты, кодирующей ЛДГ в присутствии углеродного сырья в условиях, достаточных для получения лактата.
18. Способ согласно варианту реализации 17, при этом указанная молекула нуклеиновой кислоты, кодирующая ЛДГ, происходит из Actinomyces viscosus, Acinonyx jubatus, Archilochus colubris, Bacillus anthracis, Bacillus caldolyticus, Bacillus coagulans, Bacillus megaterium, Bacillus stearothermophilus (Q9p4b6) (также известного как Geobacillus stearothermophilus), Bacillus subtilis, Bacillus thuringiensis, Bacteroides pectinophilus, Bifidobacterium longum, Bos taurus, Canis familiaris, Canis lupus, Deinococcus radiodurans, Enterococcus faecalis, Enterococcus faecium, Equus ferus, Felis catus, Kluyveromyces lactis, Kluyveromyces maxxianus, Lactobacillus acidophilus, Lactobacillus bulgaricus, Lactobacillus casei, Lactobacillus coryniformis sp.torquens, Lactobacillus delbrueckii (в том числе подвид bulgaricus), Lactobacillus fermentum, Lactobacillus helveticus, Lactobacillus johnsonii, Lactobacillus pentosus, Lactobacillus plantarum, Lactobacillus rhamnosus, Lactococcus lactis, Listeria monocytogenes, Plasmodium falciparum, Plasmodium ovale, Thermus thermophilus, Mus musculus, Oryctolagus cuniculus, Pediococcus acidilactici, Taeniopygia guttata, Rattus norvegicus, Rhizopus oryzae, Staphylococcus aureus или Streptococcus bovis.
19. Способ согласно варианту реализации 17, отличающийся тем, что указанная последовательность экзогенной нуклеиновой кислоты, кодирующая ЛДГ, кодон-оптимизирована для экспрессии в метанотрофной бактерии.
20. Способ согласно варианту реализации 17, при этом указанная молекула экзогенной нуклеиновой кислоты кодирует аминокислотную последовательность, соответствующую представленной в любой из последовательностей SEQ ID NO: 2, 4, 6, 8, 10, 60, 61, 62, 63 или 12.
21. Способ согласно варианту реализации 17, отличающийся тем, что указанная экзогенная нуклеиновая кислота, кодирующая ЛДГ, имеет последовательность, соответствующую представленной в любой из последовательностей SEQ ID NO: 1, 3, 5, 7, 9 или 11.
22. Способ согласно варианту реализации 17, отличающийся тем, что указанная экзогенная нуклеиновая кислота, кодирующая ЛДГ, функционально связана с последовательностью контроля экспрессии.
23. Способ согласно варианту реализации 22, отличающийся тем, что указанная последовательность контроля экспрессии представляет собой промотор, выбранный из пируватдекарбоксилазного промотора, метанолдегидрогеназного промотора, гексулозо-6-фосфатсинтазного промотора, промотора рибосомального белка S16, серингидроксиметилтрансферазного промотора, серинглиоксилатаминотрансферазного промотора, фосфоенолпируваткарбоксилазного промотора, Т5-промотора или Trc-промотора.
24. Способ согласно варианту реализации 17, отличающийся тем, что указанный микроорганизм обладает минимальной, недетектируемой или нокаутированной эндогенной пируватдекарбоксилазной активностью.
25. Способ согласно варианту реализации 17, отличающийся тем, что указанный микроорганизм содержит один или большее количество эндогенных ферментов метилглиоксалевого обходного пути, который(ые) обладает(ют) минимальной активностью или нокаутирован(ы).
26. Способ согласно варианту реализации 17, отличающийся тем, что указанный микроорганизм обладает минимальной, недетектируемой или нокаутированной эндогенной алкогольдегидрогеназной активностью.
27. Способ согласно варианту реализации 17, отличающийся тем, что указанный микроорганизм обладает пониженной или нокаутированной активностью эндогенной пируватдекарбоксилазы, алкогольдегидрогеназы, метилглиоксальсинтазы или любой их комбинации.
28. Способ согласно варианту реализации 17, отличающийся тем, что указанный микроорганизм способен продуцировать от приблизительно 1 мг/л до приблизительно 500 г/л лактата.
29. Способ согласно любому из вариантов реализации 17-28, при этом указанный С1-метаболизирующий микроорганизм представляет собой метанотрофный микроорганизм.
30. Способ согласно варианту реализации 29, при этом указанный метанотроф представляет собой Methylomonas, Methylobacter, Methylococcus, Methylosinus, Methylocystis, Methylomicrobium, Methanomonas, Methylocella или Methylocapsa.
31. Способ согласно варианту реализации 29, при этом указанный метанотроф представляет собой Methylococcus capsulatus, шт. Bath, Methylomonas methanica 16a (ATCC РТА 2402), Methylosinus trichosporium OB3b (NRRL B-11196), Methylosinus sporium (NRRL B-11197), Methylocystis parvus (NRRL B-11198), Methylomonas methanica (NRRL B-11199), Methylomonas albus (NRRL B-11200), Methylobacter capsulatus (NRRL B-11201), Methylobacterium organophilum (ATCC 27886), Methylomonas sp AJ-3670 (FERM P-2400), Methylocella silvestris, Methylocella palustris (ATCC 700799), Methylocella tundrae, Methylocystis daltona, штамм SB2, Methylocystis bryophila, Methylocapsa aurea KYG, Methylacidiphilum infernorum, Methylacidiphilum fumariolicum, Methyloacida kamchatkensis, Methylibium petroleiphilum или Methylomicrobium alcaliphilum.
32. Способ согласно любому из вариантов реализации 17-31, отличающийся тем, что углеродное сырье представляет собой метан, метанол, сингаз, природный газ или природный газ нетипичного происхождения.
33. Лактатная композиция, при этом δ13С лактата составляет менее чем приблизительно - 30‰.
34. Лактатная композиция согласно варианту реализации 33, при этом δ13С лактата варьирует от приблизительно - 70‰ до приблизительно -30‰.
35. Лактатная композиция согласно варианту реализации 33, при этом δ13С лактата варьирует от приблизительно - 60‰ до приблизительно - 40‰.
36. Лактатная композиция согласно любому из вариантов реализации 33-35, при этом указанную лактатную композицию продуцирует не встречающийся в природе С1-метаболизирующий микроорганизм, содержащий экзогенную нуклеиновую кислоту, кодирующую лактатдегидрогеназу (ЛДГ).
37. Лактатная композиция согласно варианту реализации 36, при этом указанный С1-метаболизирующий микроорганизм представляет собой метанотрофный микроорганизм.
38. Лактатная композиция согласно варианту реализации 37, при этом указанный метанотроф представляет собой Methylomonas, Methylobacter, Methylococcus, Methylosinus, Methylocystis, Methylomicrobium, Methanomonas, Methylocella или Methylocapsa.
39. Лактатная композиция согласно варианту реализации 37, при этом указанный метанотроф представляет собой Methylococcus capsulatus, шт. Bath, Methylomonas methanica 16a (ATCC РТА 2402), Methylosinus trichosporium OB3b (NRRL B-11196), Methylosinus sporium (NRRL B-11197), Methylocystis parvus (NRRL B-11198), Methylomonas methanica (NRRL B-11199), Methylomonas albus (NRRL B-11200), Methylobacter capsulatus (NRRL B-11201), Methylobacterium organophilum (ATCC 27886), Methylomonas sp AJ-3670 (FERM P-2400), Methylocella silvestris, Methylocella palustris (ATCC 700799), Methylocella tundrae, Methylocystis daltona, штамм SB2, Methylocystis bryophila, Methylocapsa aurea KYG, Methylacidiphilum infernorum, Methylacidiphilum fumariolicum, Methyloacida kamchatkensis, Methylibium petroleiphilum или Methylomicrobium alcaliphilum.
[0113] Лучшее понимание вышеизложенных и других аспектов настоящего изобретения может быть достигнуто при изучении приводимых ниже неограничивающих примеров.
ПРИМЕРЫ
ПРИМЕР 1
C1-МЕТАБОЛИЗИРУЮЩИЕ МИКРООРГАНИЗМЫ, СКОНСТРУИРОВАННЫЕ ДЛЯ ПОЛУЧЕНИЯ ЛАКТАТА
I. Methylococcus capsulatus Bath, сконструированные для получения лактата.
[0114] Конструировали клетки-хозяева (Methylococcus capsulatus Bath) таким образом, чтобы они содержали экзогенную нуклеиновую кислоту L-лактатдегидрогеназы (ldh), обеспечивающую продуцирование L-лактата из C1-субстрата (метана). Кодон-оптимизировали для Methylococcus capsulatus Bath и синтезировали последовательности нуклеиновых кислот, кодирующие лактатдегидрогеназы. Последовательности указанных кодон-оптимизированных нуклеиновых кислот соответствовали следующим последовательностям SEQ ID NO: 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43 и 45. Они кодируют, соответственно, следующие ЛДГ: SEQ ID NO: 2 (Streptococcus pasteurianus), 4 (Lactobacillus helveticus), 6 (Bos taurus), 8 (Pediococcus acidilactici), 10 (Rhizopus oryzae), 12 (Enterococcus faecalis), 14 (Lactobacillus casei), 16 (Bacillus megaterium), 18 (Taeniopygia guttata), 20 (Lactobacillus plantarum), 22 (Lactobaillus acidophilus), 24 (Staphylococcus aureus), 26 (Bacillus caldolyticus), 28 (Actinomyces viscosus), 30 (Bacillus anthracis), 32 (Ruminococcus torques), 34 (Listeria marthii), 36 (Bacillus subtilis), 38 (Enterococcus faecium), 40 (Bacillus thuringiensis), 42 (Geobacillus stearothermophilus), 44 (Deinococcus radiodurans), 46 (Plasmodium ovale (вариант)) и 48 (Thermus thermophilus). Нуклеиновые кислоты ЛДГ клонировали в плазмиду pMS3 (изображенную на фиг. 3) в 3'-направлении от системы промотора, представляющей собой либо промотор метанолдегидрогеназы (МДГ) с сайтом связывания рибосомы (SEQ ID NO: 49, предполагаемый промотор белка метанолдегидрогеназы М. capsulatus Bath, большая субъединица n229 (МСА0779)) (вектор pMS3×1), либо, как вариант, в тот же вектор с одним из двух разных мутированных сайтов связывания рибосомы (векторы pMS3×2 и pMS3×3), или в 3'-направлении от промотора МДГ в системе IPTG-индуцируемого (LacIq) промотора (с нативным сайтом связывания рибосомы (вектор pMS3z1), либо, как вариант, в тот же вектор с одним из двух разных мутированных сайтов связывания рибосомы (векторы pMS3z2 и pMS3z3). pMS3, происходящий из плазмиды RK2 (см. источник: Schmidhauser, et al., J. Bacteriol. 164(1): 446-55 (1985), включенный в настоящий документ посредством ссылки), представляет собой минимальную плазмиду, содержащую последовательности, которые кодируют белок инициации репликации (trfA) и промотор (Pbla), точку начала репликации (oriV), точку начала переноса (oriT), ген и промотор (промотор KanR) гена устойчивости к канамицину. Векторы вводили в Methylococcus capsulatus Bath посредством конъюгативного скрещивания на основе способов, описанных Ali и Murrell (Microbiology 155:761, 2009).
[0115] Сначала векторами трансформировали Е. coli S17-1 с применением стандартных методов электропорации. Трансформацию подтверждали путем отбора устойчивых к канамицину колоний на LB-агаре, содержащем 30 мкг/мл канамицина. Последовательность донорной плазмиды проверяли посредством секвенирования. Трансформированные колонии инокулировали в среду LB, содержащую 30 мкг/мл канамицина, и встряхивали в течение ночи при 37°C. Аликвоты (например, 100 мкл) ночных культур использовали для инокуляции свежей среды LB, содержащей 30 мкг/мл канамицина, а затем культивировали до оптической плотности (OD600) от 0,45 до 0,6 (середина логарифмической фазы роста). Аликвоты указанной второй культуры с OD, эквивалентной 1,6 (например, 3 мл культуры с OD600, составляющей 0,5) затем осаждали с помощью центрифугирования и троекратно промывали стерильной MM-W1 с помощью центрифугирования и ресуспендирования. Затем аликвоту ночной культуры объемом 10 мл помещали на стерильный нитроцеллюлозный фильтр размером 47 мм (с размером пор 0,2 мм). Донорные клетки Е. coli промывали на фильтре 50 мл стерильной среды NSM для удаления остаточной среды и антибиотика.
[0116] Параллельно образцом реципиентного штамма М. capsulatus Bath (NCIMB 11132) инокулировали флаконы для сыворотки объемом 100 мл, содержащие 20-50 мл среды MM-W1. Флаконы закрывали крышками с бутилкаучуковой прокладкой, обжимали, а затем в запечатанный флакон вводили 40-60 мл метана. Флаконы непрерывно встряхивали в термостате с температурой 42°C до достижения OD600, составляющей приблизительно 0,3. Затем с помощью центрифугирования осаждали приблизительно 5 мл культуры М. capsulatus Bath и смешивали с донорными клетками Е. coli. Указанную смесь помещали на чашку с агаром MM-W1, содержащим 0,5% дрожжевого экстракта, и инкубировали в течение 48 часов при 37°C в присутствии смеси метана и воздуха 1:1. Через 24 часа клетки ресуспендировали в 0,5 мл стерильной среды (MM-W1), и аликвоты (100 мкл) распределяли в чашках с агаром MM-W1, содержащим 7,5 мкг/мл канамицина.
[0117] Чашки инкубировали в герметичных камерах, содержащих смесь метана и воздуха 1:1, поддерживая температуру 42°C (М. capsulatus Bath). Смесь газов добавляли приблизительно каждые 2 дня до формирования колоний, как правило, через 5-8 дней. Колонии методом штриха распределяли по чашкам с NSM, содержащей канамицин, для подтверждения устойчивости к канамицину, а также для дальнейшего выделения трансформированных метанотрофных клеток от оставшихся донорных клеток Е. coli.
[0118] Экспрессию ldh или присутствие функциональной ЛДГ подтверждали посредством (1) ПЦР и секвенирования и/или (2) анализа на присутствие лактата. Например, для подтверждения переноса материал колоний кипятили при 98°C и проводили ПЦР в стандартных условиях (98°C в течение 1 мин; 30 циклов при 98°C в течение 10 с, 55°C в течение 30 секунд и 72°C в течение 1 мин; 72°C в течение 10 мин). Необязательно в качестве дополнительного контроля Е. coli XL1-Blue MRF' Kan (Stratagene, Ла-Хойя, Калифорния) могут быть снова трансформированы 1 мкл каждой из выделенных плазмид, и указанные плазмиды могут быть выделены для верификации по продуктам расщепления рестрикционными эндонуклеазами.
[0119] Рекомбинантный М. capsulatus Bath культивировали при 42°C в 24-луночных планшетах со средой MM-W1, помещенных в герметично закрытые камеры. В атмосфере свободного пространства отношение метан : воздух доводили до 1:1 по объему. Флаконы встряхивали со скоростью 200-250 об/мин. Как вариант, культуру поддерживали на планшетах со средой MM-W1, отвержденной 1,5% (масса/объем) агара, в газонепроницаемой камере, содержащей смесь газов 1:1 метан:воздух (по объему). Планшеты инкубировали в перевернутом виде в камере при 42°C.
Получение лактата из C1-субстрата (СН4)
[0120] М. capsulatus Bath трансформировали только вектором (т.е. отрицательным контролем без экзогенной кодирующей ЛДГ нуклеиновой кислоты и промотор) или вектором, содержащим экзогенную нуклеиновую кислоту ЛДГ; использовали систему с промотором для инокуляции 24-луночных планшетов с 2,5 мл среды MM-W1, содержащей 7,5 мкг/мл канамицина, на лунку. Использовали среду MM-W1 следующего состава: 0,8 мМ MgSO4 * 7H2O, 10 мМ NaNO3, 0,14 мМ CaCl2, 1,2 мМ NaHCO3, 2,35 мМ KH2PO4, 3,4 мМ K2HPO4, 20,7 мкМ Na2MoO4 * 2H2O, 1 мкМ CuSO4 * 5H2O, 10 мкМ FeIII-Na-EDTA и 1 мл/л раствора следовых металлов (содержащего 500 мг FeSO4 * 7H2O, 400 мг ZnSO4 * 7H2O, 20 мг MnCl2 * 7H2O, 50 мг CoCl2 * 6H2O, 10 мг NiCl2 * 6H2O, 15 мг H3BO3, 250 мг EDTA на литр). Фосфат, бикарбонат и FeIII-Na-EDTA добавляли после охлаждения и автоклавирования среды. Планшеты помещали в герметично закрытые камеры и свободное пространство продували смесью 1:1 воздуха и метана в качестве источника углерода для М. capsulatus Bath; планшеты запечатывали, после чего непрерывно встряхивали со скоростью 200-250 об/мин во время инкубирования при 42-45°C в течение 24 часов предварительного культивирования. Затем 0,25 мл предварительной культуры инокулировали новые 24-луночные планшеты, содержащие 2,5 мл свежей MM-W1 и канамицин, и инкубировали при 42-45°C в течение 72 часов. М. capsulatus, шт. Bath, содержащий промотор МДГ в системе IPTG-индуцируемого (LacIq) промотора, культивировали в присутствии 0,1-10 мМ IPTG). Клетки собирали центрифугированием, и анализировали супернатанты с применением набора для анализа L-лактата EnzyFluo™ в соответствии с инструкциями изготовителя (BioAssay Systems, Хейворд, Калифорния 94545, Кат. № EFLLC-100) за исключением того, что супернатанты инкубировали с реагентами набора в течение двух часов перед измерением флуоресценции. Поскольку метан был единственным доступным для клеток источником углерода, весь продуцированный лактат должен происходить из метана
Результаты
[0121] Результаты в таблице 1 соответствуют получению лактата с применением различных экзогенных нуклеиновых кислот, кодирующих лактатдегидрогеназу. В определенных случаях при синтезе лактата за счет конститутивной экспрессии ЛДГ для рекомбинантного М. capsulatus Bath наблюдалось высокое отношение лактат/OD600 (см. фиг. 1).
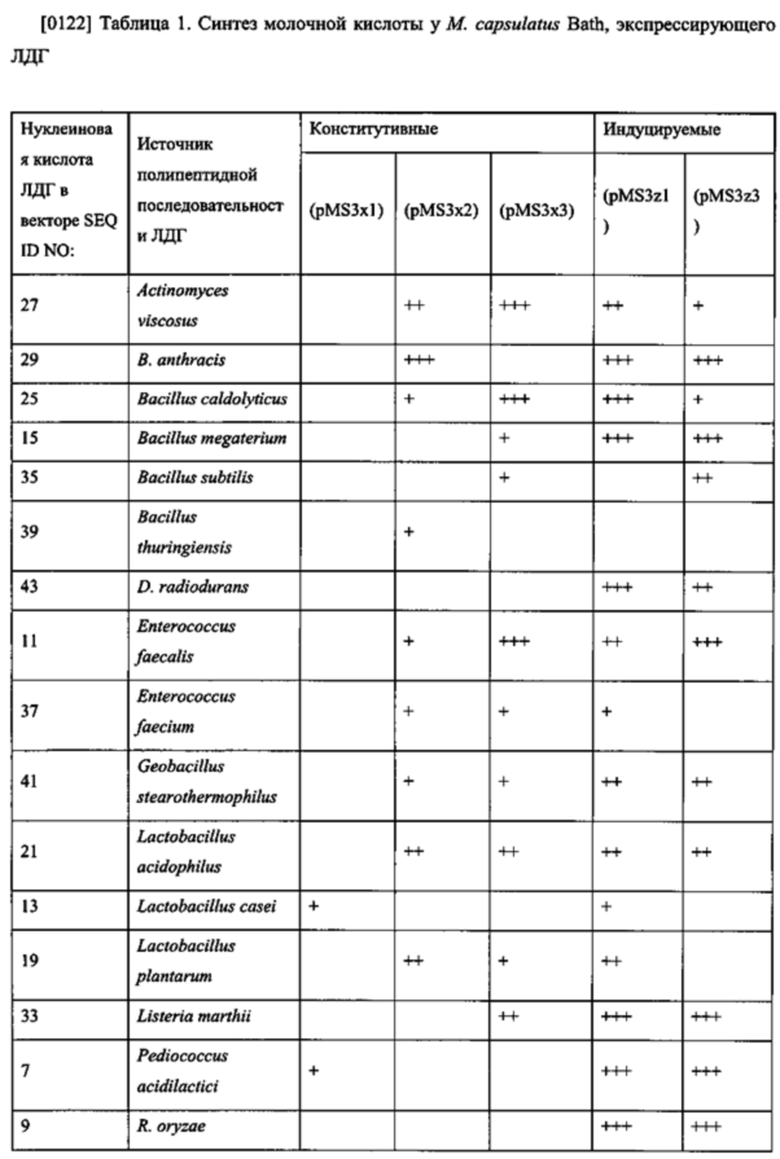
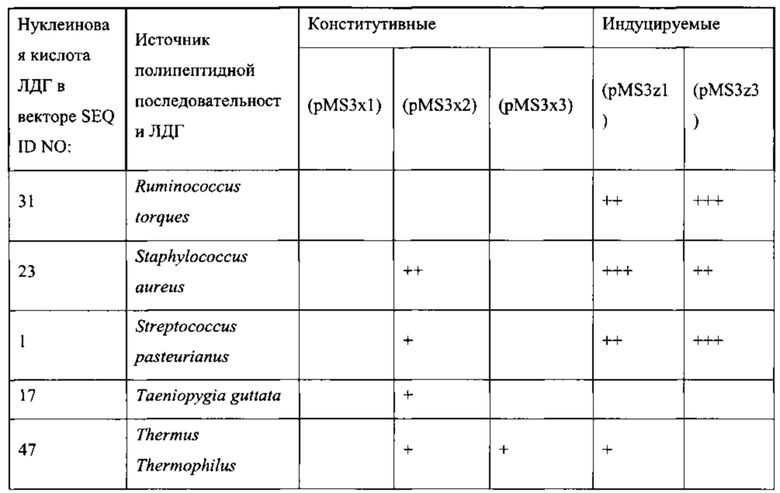
[0123] Для каждой последовательности в таблице 1 приведено максимальное детектированное значение в соответствии со следующей шкалой: +3 - 100 мкМ, ++100 - 1000 мкМ, +++ >1000 мкМ. В качестве нижнего предела определения рекомбинантных клеток как обладающих положительной активностью ЛДГ (3 мкМ) брали концентрации лактата, в 3 раза превышающие фоновую концентрацию при анализе для штаммов, по имеющимся сведениям не содержащих функциональную нуклеиновую кислоту ЛДГ (т.е. штаммов для отрицательного контроля). В ряде случаев активность ЛДГ (т.е. продуцирование лактата) выше указанного порога не детектировалась для нуклеиновых кислот, перечисленных в приведенной выше таблице, с определенными промоторами, или нуклеиновых кислот, кодирующих ЛДГ Bos Taurus (SEQ ID NO: 5), L. helveticus (SEQ ID NO: 3) или Plasmodium ovale (SEQ ID NO: 45). В трех случаях, когда экзогенная нуклеиновая кислота ЛДГ под контролем конститутивного промотора была конъюгирована в штамм-хозяина (т.е. нуклеиновые кислоты, кодирующие ЛДГ Bacillus anthracis (n:29), ЛДГ Bacillus thuringensis (SEQ ID NO: 39) и ЛДГ Deinococcus radiodurans (SEQ ID NO: 43)), колоний не наблюдалось, однако колонии все же формировались, если те же экзогенные нуклеиновые кислоты ЛДГ помещали под контроль индуцируемого промотора, что указывает на потенциальный токсический эффект лактата на клетки. Без связи с какой-либо теорией предполагается, что ввиду высокой токсичности лактата активность ЛДГ индуцирует значительное давление отбора на экспрессирующие клетки, способствующее инактивации нуклеиновой кислоты ЛДГ за счет мутаций или иных причин. Соответственно, отсутствие активности, превышающей пороговую, может быть обусловлено рядом факторов (которые наблюдались у нескольких клонов, идентифицированных как имеющие активность ниже порога детекции), в том числе мутациями нуклеиновой кислоты ЛДГ или утратой плазмиды, и в указанном случае не дает убедительных доказательств присутствия или отсутствия функциональной ЛДГ.
II. Methylosinus trichosporium OB3b и Methylomicrobium buryatense 5G, сконструированные для продуцирования лактата.
[0124] Клетки-хозяева (Methylosinus trichosporium OB3b и Methylomicrobium buryatense 5G) конструировали таким образом, что они содержали экзогенную нуклеиновую кислоту лактатдегидрогеназы (ldh) для обеспечения продуцирования L-лактата из C1-субстрата (метана). Экзогенные кодирующие ЛДГ молекулы нуклеиновых кислот из раздела I выше индивидуально клонировали в экспрессионный вектор pMS10 I (эквивалентная PMS3 плазмида с геном устойчивости к канамицину из другого источника) в 3'-направлении от системы промотора для конъюгации в М. trichosporium OB3b или М. buryatense 5G с применением способов на основе описанных Ali и Murrell (Microbiology 155:161, 2009). Для трансформации Methylosinus trichosporium OB3b в качестве промотора использовали промотор Ob3b sga (серин-глиоксилат-трансаминазы) (SEQ ID NO: 50). Для трансформации Methylomicrobium buryatense 5G в качестве промотора использовали либо промотор Methylomonas 16а moxF (метанолдегидрогеназы) (SEQ ID NO: 51), либо промотор Methylomonas 16а hps (гексулозо-6-фосфатсинтетазы) (SEQ ID NO:52).
[0125] Вкратце, мобилизуемой плазмидой, содержащей одну или большее количество представляющих интерес нуклеиновых кислот (например, ldh) и кодирующей устойчивость к канамицину, сначала трансформировали Е. coli S17-1 с применением стандартных методов электропорации. Трансформацию подтверждали путем отбора устойчивых к канамицину колоний на LB-агаре, содержащие 30 мкг/мл канамицин. Трансформированные колонии инокулировали в среду LB, содержащую 30 мкг/мл канамицина, и встряхивали при 37°C до достижения OD600 1,0. Аликвоту культуры объемом 1,0 мл помещали в стерильную пробирку Эппендорф (объемом 1,6 мл). Донорные клетки Е. coli промывали 2×1,0 мл стерильной среды MM-W1 (OB3b) или NMS (5G) для удаления остаточной среды и антибиотика.
[0126] Параллельно образцом реципиентного штамма М. trichosporium OB3b (NCIMB 11131) или М. buryatense 5G (от д-ра Мэри Лид стрем (Mary Lidstrom), Вашингтонский университет) инокулировали 100 мл флаконы для сыворотки, содержащие 20-50 мл среды MM-W1 (OB3b) или NMS (5G). Затем свободное пространство во флаконах продували смесью 1:1 кислорода и метана, и флаконы закрывали крышками с бутилкаучуковой прокладкой и обжимали. Флаконы непрерывно встряхивали в термостате при 30°C до достижения OD600, составляющей приблизительно 0,5. Затем клетки OB3b или 5G собирали центрифугированием и промывали 50 мл стерильной среды MM-W1 или NMS. Отмытые клетки ресуспендировали в стерильной среде MM-W1 или NMS до OD600 1,0 и аликвоты смешивали с донорными Е. coli в соотношении реципиент : донор, составляющем 2:1. Смесь клеток осаждали центрифугированием, клеточный осадок наносили на чашку с агаром MM-W1 или NMS, содержащим 0,5% дрожжевого экстракта, и инкубировали в течение 48 часов при 30°C в присутствии смеси 1:1 метана и воздуха. Через 48 часов клетки ресуспендировали в 1,0 мл стерильной среды и аликвоты (100 мкл) распределяли в чашках с агаром MM-W1 или NMS, содержащим 4-7,5 мкг/мл канамицина.
[0127] Чашки инкубировали в герметичных камерах, содержащих смесь 1:1 метана и воздуха, поддерживая температуру 30°C. Смесь газов добавляли каждые 2 дня до формирования колоний, как правило, через 7-14 дней. Колонии методом штриха распределяли по чашкам с MM-W1 или NMS, содержащих канамицин, для подтверждения устойчивости к канамицину, а также для дальнейшего выделения трансформированных метанотрофных клеток от оставшихся донорных клеток Е. coli.
[0128] Экспрессию ldh или присутствие функциональной ЛДГ подтверждали каким-либо одним или несколькими способами из (1) ПЦР и секвенирования, (2) вестерн-блоттинга и (3) анализа на присутствие лактата. Например, для подтверждения переноса выделяли плазмидную ДНК и проводили ПЦР с применением реакционной смеси OneTaq 2х Master Mix со стандартным буфером (New England BioLabs) в стандартных условиях (95°C в течение 5 мин; 25 циклов при 95°C в течение 30 секунд, 60°C в течение 30 секунд, и 72°C в течение 1 мин; 72°C в течение 10 мин) и наборов праймеров, специально разработанных для связывания вне и внутри нуклеиновой кислоты ldh. Исходную плазмидную ДНК, содержащую клонированную(ые) нуклеиновую(ые) кислоту(ы) ldh, использовали в качестве положительного контроля для ПЦР.
[0129] Рекомбинантный М. trichosporium OB3b или М. buryatense 5G культивировали при 30°C во флаконах для сыворотки, содержащих MM-W1 или NMS, с 4-7,5 мкг/мл канамициновой среды. В атмосфере свободного пространства отношение метан : воздух доводили до 1:1 по объему. Флаконы встряхивали со скоростью 200-250 об/мин. Как вариант, культуру поддерживали на чашках со средой MM-W1 или NMS, отвержденной 1,5% агара (масса/объем), в газонепроницаемой камере, содержащей смесь газов 1:1 метан : воздух (по объему). Чашки инкубировали в перевернутом виде в камере при 30°C.
Получение лактата из C1-субстрата (CH4)
[0130] М. trichosporium OB3b или М. buryatense 5G, трансформированные только вектором или вектором, содержащим нуклеиновую кислоту ldh, использовали для инокуляции 2,0 мл среды MM-W1 или NMS в пробирках Балча (Bellco Glass) с 5 мкг/мл канамицина. Использовали среду MM-W1 следующего состава: 0,8 мМ MgSO4 * 7H2O, 10 мМ NaNO3, 0,14 мМ CaCl2, 1,2 мМ NaHCO3, 2,35 мМ KH2PO4, 3,4 мМ K2HPO4, 20,7 мкМ Na2MoO4 * 2H2O, 1 мкМ CuSO4 * 5H2O, 10 мкМ FeIII-Na-EDTA и 1 мл/л раствора следовых металлов (содержащего на литр 500 мг FeSO4 * 7H2O, 400 мг ZnSO4 * 7H2O, 20 мг MnCl2 * 7H2O, 50 мг CoCl2 * 6H2O, 10 мг NiCl2 * 6H2O, 15 мг H3BO3, 250 мг EDTA). Фосфат, бикарбонат и FeIII-Na-EDTA добавляли после автоклавирования и охлаждения среды. Использовали среду NMS следующего состава: 1,00 г/л MgSO4 * 7H2O, 0,02 г/л CaCl2 * 6H2O, 1,00 г/л KNO3, 15 г/л NaCl, 20 мл фосфатного буфера (5,44 г/л KH2PO4, 14,34 г/л Na2HPO4 * 12H2O), 50 мл карбонатного буфера (45 мл 1 М NaHCO3 + 5 мл 1 М Na2CO3), 2 мл раствора следовых элементов (0,5 г/л Na2-EDTA, 1,0 г/л FeSO4 * 7H2O, 0,75 г/л Fe-EDTA, 0,8 г/л ZnSO4 * 7H2O, 0,005 г/л MnCl2*4H2O, 0,03 г/л Н3ВО3, 0,05 г/л CoCl2 * 6H2O, 0,4 г/л Cu-EDTA, 0,6 г/л CuCl2 * 2H2O, 0,002 г/л NiCl2 * 6H2O, 0,05 г/л Na2MoO2*2H2O) (Ojala, D.S., et al., Methods in Enzymology, Vol. 495, pp. 99-118). Стерильные фосфатный и карбонатный буферы добавляли после охлаждения раствора до комнатной температуры. Свободное пространство продували смесью 1:1 кислорода и метана в качестве источника углерода для штаммов, пробирки закрывали крышками с бутилкаучуковой прокладкой, обжимали и затем непрерывно встряхивали со скоростью 200-250 об/мин во время инкубирования при 30°C в течение 72 часов. Клетки собирали центрифугированием, и анализировали супернатанты с применением набора для анализа L-лактата EnzyFluo™ в соответствии с инструкциями изготовителя (BioAssay Systems, Хейворд, Калифорния 94545, Кат. № EFLLC-100) за исключением того, что супернатанты инкубировали с реагентами набора в течение двух часов перед измерением флуоресценции. Результаты нормировали по значениям OD600 для каждой соответствующей культуры. Поскольку метан был единственным доступным для клеток источником углерода, весь продуцированный лактат должен происходить из метана.
Результаты
[0131] М. trichosporium OB3b и М. buryatense 5G были изменены таким образом, чтобы продуцировать L-лактат, путем введения и экспрессии экзогенной нуклеиновой кислоты L-лактатдегидрогеназы. Различные экзогенные нуклеиновые кислоты лактатдегидрогеназы были функционально связаны с конститутивным промотором в экспрессионном векторе, функционирующем у метанотрофов.
[0132] Во всех тех случаях, когда синтезировался лактат, OD600 культур варьировала от 0,31 до 0,71, и все рекомбинантные штаммы продуцировали низкие, но детектируемые уровни лактата (концентрация лактата на 1-20 мкМ выше 3-кратных концентраций для штаммов, где по имеющимся данным отсутствовала функциональная нуклеиновая кислота ЛДГ).
ПРИМЕР 2
СООТНОШЕНИЕ СТАБИЛЬНЫХ ИЗОТОПОВ УГЛЕРОДА В ПРОДУКТАХ, ПРОИСХОДЯЩИХ ИЗ С1-МЕТАБОЛИЗИРУЮЩИХ МИКРООРГАНИЗМОВ
[0133] Полученную из метана молочная кислота, продуцированную сконструированными штаммами М. capsulatus Bath, анализировали на содержание углерода (% сухой массы) и отношение стабильных изотопов углерода (13С) посредством элементного анализа/проточной масс-спектрометрии изотопных отношений с применением элементного CHNOS-анализатора (vario ISOTOPE cube, Elementar, Ханау, Германия), в сочетании с масс-спектрометром для определения отношений стабильных изотопов IsoPrime100 (Isoprime, Чидл, Великобритания). Образцы биомассы метанотрофов, культивированных в ферментерах или флаконах для сыворотки, центрифугировали, ресуспендировали в деионизированной воде; объемы, соответствующие 0,2-2 мг углерода (приблизительно 0,5-5 мг сухой клеточной массы) переносили в оловянные капсулы размером 5×9 мм (Costech Analytical Technologies, Inc., Валенсия, Калифорния) и высушивали при 80°C в течение по меньшей мере 24 часов. Образцы молочной кислоты (приблизительно 0,3-1 мг), выделенной из культур, сконструированного М. capsulatus Bath, экспрессирующих кодирующие последовательности нуклеиновых кислот ЛДГ, кодирующие различные ЛДГ, аналогичным образом ресуспендировали в деионизированной воде, переносили в оловянные капсулы размером 5x9 мм и высушивали при 80°C в течение по меньшей мере 24 часов. Стандарты, содержащие по меньшей мере 0,1 мг углерода, обеспечивали достоверные значения δ13С.
[0134] Отношение изотопов обозначают знаком «дельта» (‰), при этом изотопный состав материала относительно состава стандарта, выраженный как отклонение в частях на миллион, задан как δ13С (или δ15N)=(RОбразца/RСтандарта-1)×1000, где R представляет собой молекулярное соотношение тяжелых и легких изотопных форм. Стандартом для углерода является «венский» белемнит формации PeeDee (V-PDB), а для азота - воздух. Рекомендованный Национальным институтом стандартов и технологий (NIST, National Institute of Standards and Technology) SRM («standard reference material)), стандартный эталонный материал) No. 1547, листья персика, использовали в качестве калибровочного стандарта. Все изотопные анализы проводили в Центре биогеохимии стабильных изотопов Калифорнийского университета (University of California, Беркли). Долгосрочная внешняя точность изотопного анализа С и N составляет 0,10‰ и 0,15‰, соответственно.
[0135] Отдельный М. capsulatus, шт. Bath, сконструированный для конститутивной экспрессии 4 разных нуклеиновых кислот ЛДГ (см. таблицу 2) культивировали на метане в 0,5 л флаконах для сыворотки, содержащих 130 мл среды MMS1.0 заданного состава с добавлением 15 мкг/мл канамицина. Штаммы инокулировали из периодических культур во флаконах для сыворотки (7% по объему), культивированных на той же среде с добавлением смеси 1:1 (по объему) метана и воздуха. Использовали среду MMS1.0 следующего состава: 0,8 мМ MgSO4 * 7H2O,30 мМ NaNO3, 0,14 мМ CaCl2, 1,2 мМ NaHCO3, 2,35 мМ KH2PO4, 3,4 мМ K2HPO4, 20,7 мкМ Na2MoO4 * 2H2O, 6 мкМ CuSO4 * 5H2O, 10 мкМ FeIII-Na-EDTA и 1 мл/л раствора следовых металлов (содержащего, на литр: 500 мг FeSO4 * 7H2O, 400 мг ZnSO4 * 7H2O, 20 мг MnCl2 * 7H2O, 50 мг CoCl2 * 6H2O, 10 мг NiCl2 * 6H2O, 15 мг H3BO3, 250 мг EDTA). Фосфат, бикарбонат и FeIII-Na-EDTA добавляли после автоклавирования и охлаждения среды. Конечный pH среды составлял 7,0±0,1. Флаконы для сыворотки инокулировали в двух повторностях, закрывали полыми резиновыми пробками и закачивали 60 мл газообразного метана (чистота 99%; класс 2,0, от Praxair, поставщик: Alliance Gas, Сан-Карлос, Калифорния) шприцом через стерильный 0,45 мкм фильтр и стерильные иглы 27G. Культуры инкубировали при 42°C на ротационном шейкере на 250 об/мин и измеряли рост через приблизительно 12-24-часовые интервалы, отбирая образцы объемом 1 мл для определения OD600. Субобразцы (0,5 мл) очищали центрифугированием и бесклеточные супернатанты анализировали на содержание L-лактата согласно описанию выше. После отбора образцов флаконы вентилировали и свободное пространство заполняли повторно 60 мл метана и 60-120 мл концентрированного кислорода (по меньшей мере 85% чистоты). Приблизительно через 72 часа извлекали последние образцы и оставшиеся культуральные объемы очищали центрифугированием (8000 об/мин, 10 минут), определяли pH (pH 6,7-6,9) и помещали супернатанты на хранение при -20°C до обработки для выделения и анализа продукта.

[0137] Из каждой из указанных культур жидкость-жидкостной экстракцией выделяли молочную кислоту и затем материал, экстрагированный из подкисленных супернатантов, анализировали на содержание L-молочной кислоты, относительный состав молочной кислоты и отношение стабильных изотопов углерода (значения δ13С; см. таблицу 3). Супернатанты культивированных на метане культур сконструированного М. capsulatus, шт. Bath, экспрессирующего кодирующие последовательности нуклеиновых кислот ЛДГ, кодирующие разные ЛДГ, очищали центрифугированием, доводили pH до 2 с применением концентрированной HCl и двукратно экстрагировали с применением равных объемов этилацетата (от Fisher, ВЭЖХ-чистоты). Фракции этилацетата из каждого образца объединяли, высушивали над безводным сульфатом натрия и затем концентрировали до сухого состояния под пониженным давлением посредством вращательного испарения при 40°C. Остатки растворяли в этилацетате и субобразцы анализировали на содержание L-молочной кислоты (набор для анализа L-лактата EnzyFluo™, кат. № EFLLC-100; BioAssay Systems, Хейворд, Калифорния 94545) и состав молочной кислоты посредством ГХ-МС.
[0138] Газовую хроматографию/масс-спектрометрию проводили на газовом хроматографе Agilent 6890, оснащенном Zebron ZB-5HT (номер по каталогу 7HG-G015-11; Phenomenex, Торранс, Калифорния) и масс-селективным детектором. Начальная температура колонки составляла 50°C, ее повышали со скоростью 20°C в минуту до 320°C и выдерживали в течение 5 мин. Температура подводящей и отводящей линий составляла 250°C; в качестве газа-носителя использовали гелий. Содержащие L-лактат образцы, выделенные жидкость-жидкостной экстракцией, перед анализом дериватизировали БСТФА (вводимый объем 1,0 мкл) следующим образом: к сухим лактат-содержащим экстрактам (содержащим 0,3-1 мг L-лактата) в ГХ-флаконах из боросиликатного стекла добавляли 400 мкл N,O-бис(триметилсилил)трифторацетамида (БСТФА), 400 мкл ацетонитрила и 100 мкл толуола. Готовили холостую пробу аналогичным получению эталонной пробы способом. Сосуды с образцами нагревали с дериватизирующими растворителями в течение 1 часа с применением термоблока при 50°C. Полученные дериватизированные образцы анализировали посредством ГХ-МС, используя описанные выше условия. Хроматограммы интегрировали и регистрировали индивидуальные пики для вычисления времени и площади. Хроматограммы образцов накладывали на хроматограмму холостой пробы для определения пиков, общих для холостой пробы и образцов, чтобы элиминировать их при последующей относительной количественной оценке. Массовые спектры каждого оставшегося на хроматограмме пика сравнивали с массовыми спектрами из базы данных для предварительной идентификации. Окончательный список аналитов и площадей пиков затем использовали для определения относительных концентраций молочной кислоты относительно всех аналитов. Для данного этапа считали, что все аналиты имеют эквивалентную общую интенсивность (по существу эквивалентный коэффициент отклика). Результаты показали, что относительное содержание молочной кислоты составляло 55-80% в зависимости от индивидуального образца (см. таблицу 3).
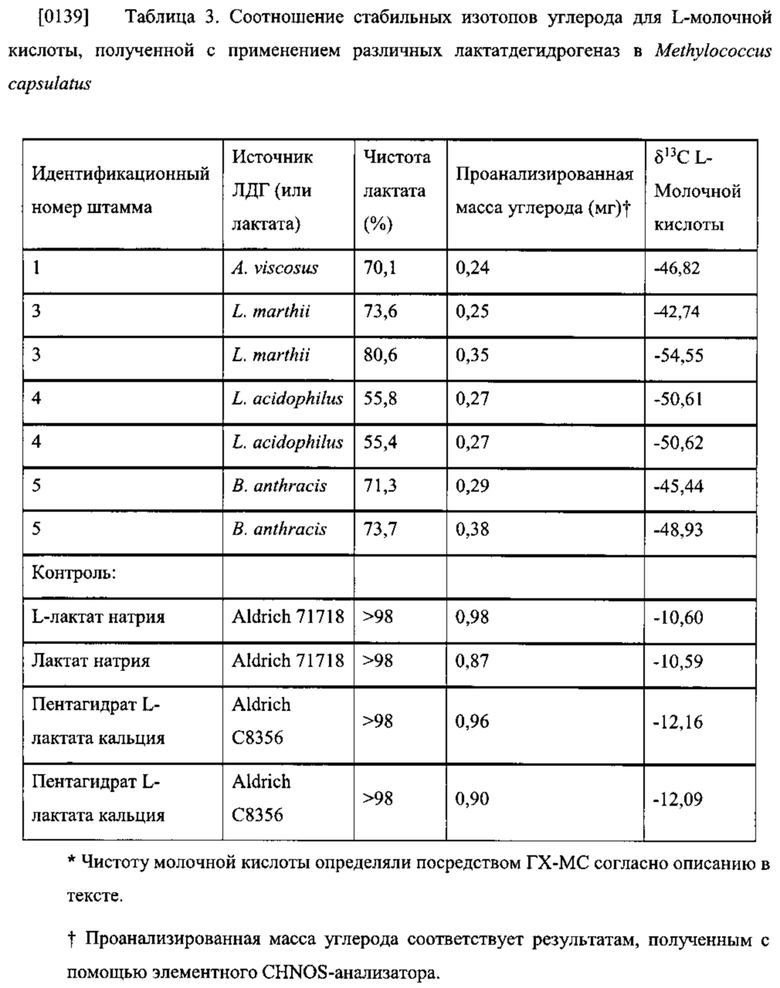
[0140] Хотя были проиллюстрированы и описаны конкретные варианты реализации настоящего изобретения, очевидно, что различные описанные выше варианты реализации могут быть скомбинированы для получения дополнительных вариантов реализации, и что могут быть произведены различные изменения без отступления от существа и объема настоящего изобретения.
[0141] Все патенты США, опубликованные заявки на патент США, заявки на патент США, иностранные патенты, заявки на иностранные патенты и непатентные публикации, упоминаемые в настоящем описании, в том числе, но не ограничиваясь перечисленными, заявка на патент США №61/836609 и заявка на патент США №61/928390, полностью включены в настоящий документ посредством ссылки. Аспекты вариантов реализации могут быть модифицированы при необходимости использования концепций из различных патентов, заявок и публикаций для получения дополнительных вариантов реализации.
[0142] Указанные и другие изменения могут быть введены во варианты реализации в свете представленного выше подробного описания. В целом, используемые в прилагаемой формуле изобретения термины не следует рассматривать как ограничивающие заявленное изобретение конкретными вариантами реализации, раскрытыми в настоящем описании и в формуле изобретения, но следует истолковывать как включающие все возможные варианты вместе с полным объемом эквивалентов, на которые распространяется формула изобретения. Соответственно, формула изобретения не ограничена настоящим описанием.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Calysta Energy, Inc./Калиста Энерджи инкорпорейтед
Silverman, Josh/Сильверман, Джош
Saville, Renee M./Сэвилл, Рене М.
Lee, Sungwon/Ли, Сонвон
Regitsky, Drew D./Режицки, Дрю Д.
Resnick, Sol M./Ресник, Сол М.
<120> КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ БИОЛОГИЧЕСКОГО ПОЛУЧЕНИЯ ЛАКТАТА ИЗ C1-СОЕДИНЕНИЙ С ПРИМЕНЕНИЕМ ТРАНСФОРМАНТОВ ЛАКТАТ ДЕГИДРОГЕНАЗЫ
<130> 200206.414WO
<150> US 61/836609
<151> 2013-06-18
<150> US 61/928390
<151> 2014-01-16
<160> 67
<170> PatentIn верс. 3.5
<210> 1
<211> 987
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Streptococcus bovis )
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(987)
<400> 1
atg acc gcg acc aag cag cac aaa aaa gtt atc ctg gtt gga gat ggg 48
Met Thr Ala Thr Lys Gln His Lys Lys Val Ile Leu Val Gly Asp Gly
1 5 10 15
gcc gtg ggc tcg tcg tat gcg ttc gcc ctc gtc aac cag ggc ata gcc 96
Ala Val Gly Ser Ser Tyr Ala Phe Ala Leu Val Asn Gln Gly Ile Ala
20 25 30
cag gaa ctg ggc atc atc gag atc ccg cag ttg ttc gac aaa gcc gtc 144
Gln Glu Leu Gly Ile Ile Glu Ile Pro Gln Leu Phe Asp Lys Ala Val
35 40 45
ggt gac gcc gag gac ctc agc cac gct ctt gcc ttc acc agc cct aaa 192
Gly Asp Ala Glu Asp Leu Ser His Ala Leu Ala Phe Thr Ser Pro Lys
50 55 60
aag atc tac gcg gca aag tac gag gat tgc gcc gac gcg gac ctg gta 240
Lys Ile Tyr Ala Ala Lys Tyr Glu Asp Cys Ala Asp Ala Asp Leu Val
65 70 75 80
gtg atc acc gcc gga gcg ccc cag aag ccc ggt gaa acg cgc ctg gac 288
Val Ile Thr Ala Gly Ala Pro Gln Lys Pro Gly Glu Thr Arg Leu Asp
85 90 95
ctg gtc ggg aag aac ttg gct atc aat aag tca att gtc acc gaa gtg 336
Leu Val Gly Lys Asn Leu Ala Ile Asn Lys Ser Ile Val Thr Glu Val
100 105 110
gtc aag tcg ggg ttc aac ggc att ttc ctc gtg gcc gcg aat cca gtg 384
Val Lys Ser Gly Phe Asn Gly Ile Phe Leu Val Ala Ala Asn Pro Val
115 120 125
gac gtg ctg aca tac agc acc tgg aag ttc tcc ggc ttc ccg aag gaa 432
Asp Val Leu Thr Tyr Ser Thr Trp Lys Phe Ser Gly Phe Pro Lys Glu
130 135 140
cgg gtc atc ggt agc ggc acg agc ctg gac tcg gcc agg ttt cgg caa 480
Arg Val Ile Gly Ser Gly Thr Ser Leu Asp Ser Ala Arg Phe Arg Gln
145 150 155 160
gcc ctg gcg gag aag ctg gat gtc gat gcc cgc tcc gtc cac gca tac 528
Ala Leu Ala Glu Lys Leu Asp Val Asp Ala Arg Ser Val His Ala Tyr
165 170 175
atc atg ggc gag cat ggc gac tcg gag ttc gcc gtg tgg agc cat gcg 576
Ile Met Gly Glu His Gly Asp Ser Glu Phe Ala Val Trp Ser His Ala
180 185 190
aat gtc gct ggg gtg aat ctg gag aac tat ctg aaa gac gtc cag aac 624
Asn Val Ala Gly Val Asn Leu Glu Asn Tyr Leu Lys Asp Val Gln Asn
195 200 205
gtg aac gaa gcg gaa ctc gtg gag ctg ttc gag ggc gtc cgt gat gcg 672
Val Asn Glu Ala Glu Leu Val Glu Leu Phe Glu Gly Val Arg Asp Ala
210 215 220
gcg tat tcc atc atc aac aaa aag ggc gcc act ttc tac ggc atc gcc 720
Ala Tyr Ser Ile Ile Asn Lys Lys Gly Ala Thr Phe Tyr Gly Ile Ala
225 230 235 240
gtg gcg ctc gcc cgg atc acg aag gcc att ttg aac gat gaa aac gcc 768
Val Ala Leu Ala Arg Ile Thr Lys Ala Ile Leu Asn Asp Glu Asn Ala
245 250 255
gtc ctg ccg ctg tcc gtg ttc cag gaa ggg cag tat ccg ggt gtg acc 816
Val Leu Pro Leu Ser Val Phe Gln Glu Gly Gln Tyr Pro Gly Val Thr
260 265 270
gac tgc tac atc ggc cag ccc gcc atc gtc gga gcc cat ggc atc gta 864
Asp Cys Tyr Ile Gly Gln Pro Ala Ile Val Gly Ala His Gly Ile Val
275 280 285
cgc ccc gtc aac atc ccg ctc aac gac gcg gag cag caa aag atg gaa 912
Arg Pro Val Asn Ile Pro Leu Asn Asp Ala Glu Gln Gln Lys Met Glu
290 295 300
gca tct gca aaa gaa ctg aag gcc atc atc gac gaa gcc ttc tcc aaa 960
Ala Ser Ala Lys Glu Leu Lys Ala Ile Ile Asp Glu Ala Phe Ser Lys
305 310 315 320
gaa gag ttt gcc agt gcg gcg aag taa 987
Glu Glu Phe Ala Ser Ala Ala Lys
325
<210> 2
<211> 328
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 2
Met Thr Ala Thr Lys Gln His Lys Lys Val Ile Leu Val Gly Asp Gly
1 5 10 15
Ala Val Gly Ser Ser Tyr Ala Phe Ala Leu Val Asn Gln Gly Ile Ala
20 25 30
Gln Glu Leu Gly Ile Ile Glu Ile Pro Gln Leu Phe Asp Lys Ala Val
35 40 45
Gly Asp Ala Glu Asp Leu Ser His Ala Leu Ala Phe Thr Ser Pro Lys
50 55 60
Lys Ile Tyr Ala Ala Lys Tyr Glu Asp Cys Ala Asp Ala Asp Leu Val
65 70 75 80
Val Ile Thr Ala Gly Ala Pro Gln Lys Pro Gly Glu Thr Arg Leu Asp
85 90 95
Leu Val Gly Lys Asn Leu Ala Ile Asn Lys Ser Ile Val Thr Glu Val
100 105 110
Val Lys Ser Gly Phe Asn Gly Ile Phe Leu Val Ala Ala Asn Pro Val
115 120 125
Asp Val Leu Thr Tyr Ser Thr Trp Lys Phe Ser Gly Phe Pro Lys Glu
130 135 140
Arg Val Ile Gly Ser Gly Thr Ser Leu Asp Ser Ala Arg Phe Arg Gln
145 150 155 160
Ala Leu Ala Glu Lys Leu Asp Val Asp Ala Arg Ser Val His Ala Tyr
165 170 175
Ile Met Gly Glu His Gly Asp Ser Glu Phe Ala Val Trp Ser His Ala
180 185 190
Asn Val Ala Gly Val Asn Leu Glu Asn Tyr Leu Lys Asp Val Gln Asn
195 200 205
Val Asn Glu Ala Glu Leu Val Glu Leu Phe Glu Gly Val Arg Asp Ala
210 215 220
Ala Tyr Ser Ile Ile Asn Lys Lys Gly Ala Thr Phe Tyr Gly Ile Ala
225 230 235 240
Val Ala Leu Ala Arg Ile Thr Lys Ala Ile Leu Asn Asp Glu Asn Ala
245 250 255
Val Leu Pro Leu Ser Val Phe Gln Glu Gly Gln Tyr Pro Gly Val Thr
260 265 270
Asp Cys Tyr Ile Gly Gln Pro Ala Ile Val Gly Ala His Gly Ile Val
275 280 285
Arg Pro Val Asn Ile Pro Leu Asn Asp Ala Glu Gln Gln Lys Met Glu
290 295 300
Ala Ser Ala Lys Glu Leu Lys Ala Ile Ile Asp Glu Ala Phe Ser Lys
305 310 315 320
Glu Glu Phe Ala Ser Ala Ala Lys
325
<210> 3
<211> 924
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Lactobacillus helveticus)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(924)
<400> 3
atg tct agg aaa gtg ttt ctt gtg ggt gat ggt gcg gtg ggg tca acg 48
Met Ser Arg Lys Val Phe Leu Val Gly Asp Gly Ala Val Gly Ser Thr
1 5 10 15
ttt gcg aac gat ttg ttg cag aat gcg aaa gtg gac gag ctc gcc atc 96
Phe Ala Asn Asp Leu Leu Gln Asn Ala Lys Val Asp Glu Leu Ala Ile
20 25 30
ttc gag gtc gcc aag gat cgg ccg gta ggc gac gcg atg gac ctg gaa 144
Phe Glu Val Ala Lys Asp Arg Pro Val Gly Asp Ala Met Asp Leu Glu
35 40 45
gat atc acc ccc ttc atg ggc cag acg gat atc cac ccg gca gac tac 192
Asp Ile Thr Pro Phe Met Gly Gln Thr Asp Ile His Pro Ala Asp Tyr
50 55 60
tcg gac gcg aaa gac gcc gac gtg tgc gtc atc gct gcg ggt gtg ccc 240
Ser Asp Ala Lys Asp Ala Asp Val Cys Val Ile Ala Ala Gly Val Pro
65 70 75 80
cgc aag ccc ggt gaa aca cgg ctc gac ctg gtc gcg aaa aac gtc aag 288
Arg Lys Pro Gly Glu Thr Arg Leu Asp Leu Val Ala Lys Asn Val Lys
85 90 95
ata ctg aag tcc atc gtg cag ccg gtc gtg gaa tcc ggc ttc aaa ggc 336
Ile Leu Lys Ser Ile Val Gln Pro Val Val Glu Ser Gly Phe Lys Gly
100 105 110
gtg ttc gtt gtg tcg gcg aac ccg gtc gac att ctg acc acc ctc acg 384
Val Phe Val Val Ser Ala Asn Pro Val Asp Ile Leu Thr Thr Leu Thr
115 120 125
cag aag ttg agt ggc ttc ccg aag aac cgc gtg atc gga acc ggc acc 432
Gln Lys Leu Ser Gly Phe Pro Lys Asn Arg Val Ile Gly Thr Gly Thr
130 135 140
agc ctg gat tcg atg cgt ctg cgc gtg gag ctc gcc aag aaa ctg aac 480
Ser Leu Asp Ser Met Arg Leu Arg Val Glu Leu Ala Lys Lys Leu Asn
145 150 155 160
gtt ccc gtc gct aag gtc aat agc atg gtc ctc gga gaa cac ggc gac 528
Val Pro Val Ala Lys Val Asn Ser Met Val Leu Gly Glu His Gly Asp
165 170 175
acg tcg ttc gag aat ttc gac gag agc acc gtg gac ggc aag ccg ctg 576
Thr Ser Phe Glu Asn Phe Asp Glu Ser Thr Val Asp Gly Lys Pro Leu
180 185 190
cgc gac tac gcc gag atc aac gac gat gtc ctg agc gag atc gaa acc 624
Arg Asp Tyr Ala Glu Ile Asn Asp Asp Val Leu Ser Glu Ile Glu Thr
195 200 205
gac gtc cga aag aaa ggc gag aag atc atc gcc aaa aag ggc gcc acg 672
Asp Val Arg Lys Lys Gly Glu Lys Ile Ile Ala Lys Lys Gly Ala Thr
210 215 220
ttc tat ggg gtc gcc atg atg ctg acc cag atc gta tcc gcc att ctg 720
Phe Tyr Gly Val Ala Met Met Leu Thr Gln Ile Val Ser Ala Ile Leu
225 230 235 240
gac aat cgg tcc atc tgc ctg ccg ctg agc gcc cct atc aac ggc gaa 768
Asp Asn Arg Ser Ile Cys Leu Pro Leu Ser Ala Pro Ile Asn Gly Glu
245 250 255
tat ggc atc aag cat gac ctc tac ctg ggc acc cca gcc gtg atc aac 816
Tyr Gly Ile Lys His Asp Leu Tyr Leu Gly Thr Pro Ala Val Ile Asn
260 265 270
ggg gaa ggg atc gaa cag gtc atc gag act aag ctg tcg gat gcc gaa 864
Gly Glu Gly Ile Glu Gln Val Ile Glu Thr Lys Leu Ser Asp Ala Glu
275 280 285
aag gcc aag atg att aac tcc gca gat aag atg caa gaa gtc ctg ggc 912
Lys Ala Lys Met Ile Asn Ser Ala Asp Lys Met Gln Glu Val Leu Gly
290 295 300
gga atc gag taa 924
Gly Ile Glu
305
<210> 4
<211> 307
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 4
Met Ser Arg Lys Val Phe Leu Val Gly Asp Gly Ala Val Gly Ser Thr
1 5 10 15
Phe Ala Asn Asp Leu Leu Gln Asn Ala Lys Val Asp Glu Leu Ala Ile
20 25 30
Phe Glu Val Ala Lys Asp Arg Pro Val Gly Asp Ala Met Asp Leu Glu
35 40 45
Asp Ile Thr Pro Phe Met Gly Gln Thr Asp Ile His Pro Ala Asp Tyr
50 55 60
Ser Asp Ala Lys Asp Ala Asp Val Cys Val Ile Ala Ala Gly Val Pro
65 70 75 80
Arg Lys Pro Gly Glu Thr Arg Leu Asp Leu Val Ala Lys Asn Val Lys
85 90 95
Ile Leu Lys Ser Ile Val Gln Pro Val Val Glu Ser Gly Phe Lys Gly
100 105 110
Val Phe Val Val Ser Ala Asn Pro Val Asp Ile Leu Thr Thr Leu Thr
115 120 125
Gln Lys Leu Ser Gly Phe Pro Lys Asn Arg Val Ile Gly Thr Gly Thr
130 135 140
Ser Leu Asp Ser Met Arg Leu Arg Val Glu Leu Ala Lys Lys Leu Asn
145 150 155 160
Val Pro Val Ala Lys Val Asn Ser Met Val Leu Gly Glu His Gly Asp
165 170 175
Thr Ser Phe Glu Asn Phe Asp Glu Ser Thr Val Asp Gly Lys Pro Leu
180 185 190
Arg Asp Tyr Ala Glu Ile Asn Asp Asp Val Leu Ser Glu Ile Glu Thr
195 200 205
Asp Val Arg Lys Lys Gly Glu Lys Ile Ile Ala Lys Lys Gly Ala Thr
210 215 220
Phe Tyr Gly Val Ala Met Met Leu Thr Gln Ile Val Ser Ala Ile Leu
225 230 235 240
Asp Asn Arg Ser Ile Cys Leu Pro Leu Ser Ala Pro Ile Asn Gly Glu
245 250 255
Tyr Gly Ile Lys His Asp Leu Tyr Leu Gly Thr Pro Ala Val Ile Asn
260 265 270
Gly Glu Gly Ile Glu Gln Val Ile Glu Thr Lys Leu Ser Asp Ala Glu
275 280 285
Lys Ala Lys Met Ile Asn Ser Ala Asp Lys Met Gln Glu Val Leu Gly
290 295 300
Gly Ile Glu
305
<210> 5
<211> 1002
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (вариант ЛДГ Bos taurus)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(1002)
<400> 5
atg gcg act ctc aaa gat cag ctc ata cag aac ttg ctt aaa gaa gaa 48
Met Ala Thr Leu Lys Asp Gln Leu Ile Gln Asn Leu Leu Lys Glu Glu
1 5 10 15
cat gtg ccc cag aac aag atc acc atc gtg ggt gtg ggc gcc gtt ggg 96
His Val Pro Gln Asn Lys Ile Thr Ile Val Gly Val Gly Ala Val Gly
20 25 30
atg gcc tgc gcg atc agc atc ctg atg aaa gat ctc gcc gac gaa gtc 144
Met Ala Cys Ala Ile Ser Ile Leu Met Lys Asp Leu Ala Asp Glu Val
35 40 45
gca ctg gtt gac gtg atg gaa gat aag ctc aag ggt aat gaa atg atg 192
Ala Leu Val Asp Val Met Glu Asp Lys Leu Lys Gly Asn Glu Met Met
50 55 60
gac ctc cag cat ggg tca ctg ttc ctg cga acc cca aag att gtc tcc 240
Asp Leu Gln His Gly Ser Leu Phe Leu Arg Thr Pro Lys Ile Val Ser
65 70 75 80
gga aag gac tac aac gtc acc gcc aac agc cgg ctg gtg atc atc acg 288
Gly Lys Asp Tyr Asn Val Thr Ala Asn Ser Arg Leu Val Ile Ile Thr
85 90 95
gct ggg gca cgc caa cag gaa ggc gag tcg agg ctg aac ctc gtc cag 336
Ala Gly Ala Arg Gln Gln Glu Gly Glu Ser Arg Leu Asn Leu Val Gln
100 105 110
cgc aat gtc aac atc ttt aag ttc atc atc ccg aac atc gtg aag tac 384
Arg Asn Val Asn Ile Phe Lys Phe Ile Ile Pro Asn Ile Val Lys Tyr
115 120 125
tcc ccg aac tgc aag ctg ttg gtt gtg tcg aat ccc gtg gat atc ctg 432
Ser Pro Asn Cys Lys Leu Leu Val Val Ser Asn Pro Val Asp Ile Leu
130 135 140
acg tac gtc gcc tgg aag att tcg ggc ttc ccc aag aac cgg gta atc 480
Thr Tyr Val Ala Trp Lys Ile Ser Gly Phe Pro Lys Asn Arg Val Ile
145 150 155 160
ggc agc ggc tgt aac ctg gat agc gcg cgt ttc cgc tac ctg atg ggc 528
Gly Ser Gly Cys Asn Leu Asp Ser Ala Arg Phe Arg Tyr Leu Met Gly
165 170 175
gag cgc ttg gga gtc cac ccg ctg agc tgc cac ggg tgg atc ctc ggt 576
Glu Arg Leu Gly Val His Pro Leu Ser Cys His Gly Trp Ile Leu Gly
180 185 190
gag cat ggc gac tcc tcg gtg cct gtc tgg tct ggc gtc aat gta gcc 624
Glu His Gly Asp Ser Ser Val Pro Val Trp Ser Gly Val Asn Val Ala
195 200 205
ggc gtc agt ctg aag aat ctg cac ccc gag ctg ggc acc gac gcc gac 672
Gly Val Ser Leu Lys Asn Leu His Pro Glu Leu Gly Thr Asp Ala Asp
210 215 220
aaa gag cag tgg aaa gcc gtc cac aag caa gtg gtg gac tcg gcg tat 720
Lys Glu Gln Trp Lys Ala Val His Lys Gln Val Val Asp Ser Ala Tyr
225 230 235 240
gaa gtc atc aag ctg aag ggc tat acc agt tgg gcg atc ggc ctg agc 768
Glu Val Ile Lys Leu Lys Gly Tyr Thr Ser Trp Ala Ile Gly Leu Ser
245 250 255
gtg gcc gac ctg gcg gag tcc att atg aag aac ctt cgg cgc gtc cac 816
Val Ala Asp Leu Ala Glu Ser Ile Met Lys Asn Leu Arg Arg Val His
260 265 270
ccg atc tcg acc atg atc aaa ggc ctc tac ggg atc aaa gaa gat gtg 864
Pro Ile Ser Thr Met Ile Lys Gly Leu Tyr Gly Ile Lys Glu Asp Val
275 280 285
ttc ctc tcc gtc ccg tgc atc ttg ggc cag aac gga atc tcc gac gtc 912
Phe Leu Ser Val Pro Cys Ile Leu Gly Gln Asn Gly Ile Ser Asp Val
290 295 300
gtg aag gtc aca ctg acg cat gaa gaa gaa gcg tgc ctg aag aag tcg 960
Val Lys Val Thr Leu Thr His Glu Glu Glu Ala Cys Leu Lys Lys Ser
305 310 315 320
gcc gac acc ctg tgg ggc atc cag aaa gaa cta cag ttc taa 1002
Ala Asp Thr Leu Trp Gly Ile Gln Lys Glu Leu Gln Phe
325 330
<210> 6
<211> 333
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 6
Met Ala Thr Leu Lys Asp Gln Leu Ile Gln Asn Leu Leu Lys Glu Glu
1 5 10 15
His Val Pro Gln Asn Lys Ile Thr Ile Val Gly Val Gly Ala Val Gly
20 25 30
Met Ala Cys Ala Ile Ser Ile Leu Met Lys Asp Leu Ala Asp Glu Val
35 40 45
Ala Leu Val Asp Val Met Glu Asp Lys Leu Lys Gly Asn Glu Met Met
50 55 60
Asp Leu Gln His Gly Ser Leu Phe Leu Arg Thr Pro Lys Ile Val Ser
65 70 75 80
Gly Lys Asp Tyr Asn Val Thr Ala Asn Ser Arg Leu Val Ile Ile Thr
85 90 95
Ala Gly Ala Arg Gln Gln Glu Gly Glu Ser Arg Leu Asn Leu Val Gln
100 105 110
Arg Asn Val Asn Ile Phe Lys Phe Ile Ile Pro Asn Ile Val Lys Tyr
115 120 125
Ser Pro Asn Cys Lys Leu Leu Val Val Ser Asn Pro Val Asp Ile Leu
130 135 140
Thr Tyr Val Ala Trp Lys Ile Ser Gly Phe Pro Lys Asn Arg Val Ile
145 150 155 160
Gly Ser Gly Cys Asn Leu Asp Ser Ala Arg Phe Arg Tyr Leu Met Gly
165 170 175
Glu Arg Leu Gly Val His Pro Leu Ser Cys His Gly Trp Ile Leu Gly
180 185 190
Glu His Gly Asp Ser Ser Val Pro Val Trp Ser Gly Val Asn Val Ala
195 200 205
Gly Val Ser Leu Lys Asn Leu His Pro Glu Leu Gly Thr Asp Ala Asp
210 215 220
Lys Glu Gln Trp Lys Ala Val His Lys Gln Val Val Asp Ser Ala Tyr
225 230 235 240
Glu Val Ile Lys Leu Lys Gly Tyr Thr Ser Trp Ala Ile Gly Leu Ser
245 250 255
Val Ala Asp Leu Ala Glu Ser Ile Met Lys Asn Leu Arg Arg Val His
260 265 270
Pro Ile Ser Thr Met Ile Lys Gly Leu Tyr Gly Ile Lys Glu Asp Val
275 280 285
Phe Leu Ser Val Pro Cys Ile Leu Gly Gln Asn Gly Ile Ser Asp Val
290 295 300
Val Lys Val Thr Leu Thr His Glu Glu Glu Ala Cys Leu Lys Lys Ser
305 310 315 320
Ala Asp Thr Leu Trp Gly Ile Gln Lys Glu Leu Gln Phe
325 330
<210> 7
<211> 260
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Pediococcus acidilactici)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(258)
<400> 7
atg tct aat att caa aat cat caa aaa gtt gtc ctc gtc ggt gac ggt 48
Met Ser Asn Ile Gln Asn His Gln Lys Val Val Leu Val Gly Asp Gly
1 5 10 15
gcc gta ggt tct agt tac gca ttc gcg atg gca caa caa gga atc gct 96
Ala Val Gly Ser Ser Tyr Ala Phe Ala Met Ala Gln Gln Gly Ile Ala
20 25 30
gaa gaa ttc gtc att gtc gac gtt gtt aag gat cgt aca gtt ggg gac 144
Glu Glu Phe Val Ile Val Asp Val Val Lys Asp Arg Thr Val Gly Asp
35 40 45
gca ttg gac ctt gaa gat gct act cca ttc aca gct cca aag aac atc 192
Ala Leu Asp Leu Glu Asp Ala Thr Pro Phe Thr Ala Pro Lys Asn Ile
50 55 60
tac tct ggt gaa tac tca gac tgc aag gat gct gac tta gtt gtt atc 240
Tyr Ser Gly Glu Tyr Ser Asp Cys Lys Asp Ala Asp Leu Val Val Ile
65 70 75 80
aca gct ggc gca cca caa aa 260
Thr Ala Gly Ala Pro Gln
85
<210> 8
<211> 86
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 8
Met Ser Asn Ile Gln Asn His Gln Lys Val Val Leu Val Gly Asp Gly
1 5 10 15
Ala Val Gly Ser Ser Tyr Ala Phe Ala Met Ala Gln Gln Gly Ile Ala
20 25 30
Glu Glu Phe Val Ile Val Asp Val Val Lys Asp Arg Thr Val Gly Asp
35 40 45
Ala Leu Asp Leu Glu Asp Ala Thr Pro Phe Thr Ala Pro Lys Asn Ile
50 55 60
Tyr Ser Gly Glu Tyr Ser Asp Cys Lys Asp Ala Asp Leu Val Val Ile
65 70 75 80
Thr Ala Gly Ala Pro Gln
85
<210> 9
<211> 963
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Rhizopus oryzae)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(963)
<400> 9
atg gta tta cac agc aaa gtc gca ata gtc gga gcg gga gca gtt ggc 48
Met Val Leu His Ser Lys Val Ala Ile Val Gly Ala Gly Ala Val Gly
1 5 10 15
gca tct aca gca tac gcg ttg atg ttc aag aac atc tgc acg gaa att 96
Ala Ser Thr Ala Tyr Ala Leu Met Phe Lys Asn Ile Cys Thr Glu Ile
20 25 30
atc gtg gtc gac gtg aac ccc gat atc gtg cag gcc cag gtg ctg gac 144
Ile Val Val Asp Val Asn Pro Asp Ile Val Gln Ala Gln Val Leu Asp
35 40 45
ctg gcc gat gcc gct agc atc tcc cac act cct atc agg gcg gga tcg 192
Leu Ala Asp Ala Ala Ser Ile Ser His Thr Pro Ile Arg Ala Gly Ser
50 55 60
gtc gaa gaa gcc ggg cag gcc gat atc gtt gtg ata acc gcg ggt gcc 240
Val Glu Glu Ala Gly Gln Ala Asp Ile Val Val Ile Thr Ala Gly Ala
65 70 75 80
aag cag cgc gag ggc gag ccg cgc acc aag ctg atc gaa cgg aat tac 288
Lys Gln Arg Glu Gly Glu Pro Arg Thr Lys Leu Ile Glu Arg Asn Tyr
85 90 95
cgg gtc ctg cag agc atc atc ggc ggc atg cag ccc atc cgt cca gac 336
Arg Val Leu Gln Ser Ile Ile Gly Gly Met Gln Pro Ile Arg Pro Asp
100 105 110
gcc gtg atc ctc gtg gtc gct aac ccg gtc gac att ctg acc cat atc 384
Ala Val Ile Leu Val Val Ala Asn Pro Val Asp Ile Leu Thr His Ile
115 120 125
gct aag acc ctc tcc ggc ctg ccg ccc aac caa gtg atc ggc tcg ggt 432
Ala Lys Thr Leu Ser Gly Leu Pro Pro Asn Gln Val Ile Gly Ser Gly
130 135 140
acc tac ctg gac acg acg cgc ctg cgc gtg cac ctc ggc gac gtg ttc 480
Thr Tyr Leu Asp Thr Thr Arg Leu Arg Val His Leu Gly Asp Val Phe
145 150 155 160
gac gtc aac ccg cag agc atc cac gcg ttc gtg ctg ggc gag cac ggc 528
Asp Val Asn Pro Gln Ser Ile His Ala Phe Val Leu Gly Glu His Gly
165 170 175
gac tcg caa atg atc gcg tgg gaa gcc gcg tcg atc ggc ggt cag ccc 576
Asp Ser Gln Met Ile Ala Trp Glu Ala Ala Ser Ile Gly Gly Gln Pro
180 185 190
ctc acg agc ttc ccg gag ttc gcg aag ctg gac aag acc gcc atc tca 624
Leu Thr Ser Phe Pro Glu Phe Ala Lys Leu Asp Lys Thr Ala Ile Ser
195 200 205
aag gcc atc tcc ggc aag gcc atg gaa atc att cgg ttg aaa ggc gcc 672
Lys Ala Ile Ser Gly Lys Ala Met Glu Ile Ile Arg Leu Lys Gly Ala
210 215 220
acc ttc tac ggg atc ggg gcg tgt gcc gcc gat ctg gtg cat acc atc 720
Thr Phe Tyr Gly Ile Gly Ala Cys Ala Ala Asp Leu Val His Thr Ile
225 230 235 240
atg ctg aac cgt aag agt gtc cat ccg gtg agt gtg tac gtc gag aaa 768
Met Leu Asn Arg Lys Ser Val His Pro Val Ser Val Tyr Val Glu Lys
245 250 255
tat ggt gcg acg ttc tcc atg cct gcg aag ctg ggc tgg cgc ggc gta 816
Tyr Gly Ala Thr Phe Ser Met Pro Ala Lys Leu Gly Trp Arg Gly Val
260 265 270
gag caa atc tat gaa gtc ccc ttg acc gaa gaa gaa gaa gcc ctt ctg 864
Glu Gln Ile Tyr Glu Val Pro Leu Thr Glu Glu Glu Glu Ala Leu Leu
275 280 285
gtc aaa tcg gtc gag gcc ctc aag agc gtc gag tat tcc agt acc aag 912
Val Lys Ser Val Glu Ala Leu Lys Ser Val Glu Tyr Ser Ser Thr Lys
290 295 300
gtg ccc gag aaa aaa gtc cat gcg acc tcg ttt agc aag tcc aat tgc 960
Val Pro Glu Lys Lys Val His Ala Thr Ser Phe Ser Lys Ser Asn Cys
305 310 315 320
taa 963
<210> 10
<211> 320
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 10
Met Val Leu His Ser Lys Val Ala Ile Val Gly Ala Gly Ala Val Gly
1 5 10 15
Ala Ser Thr Ala Tyr Ala Leu Met Phe Lys Asn Ile Cys Thr Glu Ile
20 25 30
Ile Val Val Asp Val Asn Pro Asp Ile Val Gln Ala Gln Val Leu Asp
35 40 45
Leu Ala Asp Ala Ala Ser Ile Ser His Thr Pro Ile Arg Ala Gly Ser
50 55 60
Val Glu Glu Ala Gly Gln Ala Asp Ile Val Val Ile Thr Ala Gly Ala
65 70 75 80
Lys Gln Arg Glu Gly Glu Pro Arg Thr Lys Leu Ile Glu Arg Asn Tyr
85 90 95
Arg Val Leu Gln Ser Ile Ile Gly Gly Met Gln Pro Ile Arg Pro Asp
100 105 110
Ala Val Ile Leu Val Val Ala Asn Pro Val Asp Ile Leu Thr His Ile
115 120 125
Ala Lys Thr Leu Ser Gly Leu Pro Pro Asn Gln Val Ile Gly Ser Gly
130 135 140
Thr Tyr Leu Asp Thr Thr Arg Leu Arg Val His Leu Gly Asp Val Phe
145 150 155 160
Asp Val Asn Pro Gln Ser Ile His Ala Phe Val Leu Gly Glu His Gly
165 170 175
Asp Ser Gln Met Ile Ala Trp Glu Ala Ala Ser Ile Gly Gly Gln Pro
180 185 190
Leu Thr Ser Phe Pro Glu Phe Ala Lys Leu Asp Lys Thr Ala Ile Ser
195 200 205
Lys Ala Ile Ser Gly Lys Ala Met Glu Ile Ile Arg Leu Lys Gly Ala
210 215 220
Thr Phe Tyr Gly Ile Gly Ala Cys Ala Ala Asp Leu Val His Thr Ile
225 230 235 240
Met Leu Asn Arg Lys Ser Val His Pro Val Ser Val Tyr Val Glu Lys
245 250 255
Tyr Gly Ala Thr Phe Ser Met Pro Ala Lys Leu Gly Trp Arg Gly Val
260 265 270
Glu Gln Ile Tyr Glu Val Pro Leu Thr Glu Glu Glu Glu Ala Leu Leu
275 280 285
Val Lys Ser Val Glu Ala Leu Lys Ser Val Glu Tyr Ser Ser Thr Lys
290 295 300
Val Pro Glu Lys Lys Val His Ala Thr Ser Phe Ser Lys Ser Asn Cys
305 310 315 320
<210> 11
<211> 984
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Enterococcus faecalis)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(984)
<400> 11
atg acc gcg gct gcc gga aat aag gac cac cag aaa gtg ata ctg gtc 48
Met Thr Ala Ala Ala Gly Asn Lys Asp His Gln Lys Val Ile Leu Val
1 5 10 15
ggg gac ggt gcc gtg ggc agc tcg tat gca ttc gcg ttg gtg acc cag 96
Gly Asp Gly Ala Val Gly Ser Ser Tyr Ala Phe Ala Leu Val Thr Gln
20 25 30
aac atc gcc caa gaa gtc ggc atc ata gat atc aac gtc ccc aag acc 144
Asn Ile Ala Gln Glu Val Gly Ile Ile Asp Ile Asn Val Pro Lys Thr
35 40 45
gag ggc gac gcc ctg gat ctc tcc cac gcc ctg gcc ttc acc agc ccc 192
Glu Gly Asp Ala Leu Asp Leu Ser His Ala Leu Ala Phe Thr Ser Pro
50 55 60
aaa aag atc tac gcg gcc acc tac gac gat tgc cat gac gcg gat ctc 240
Lys Lys Ile Tyr Ala Ala Thr Tyr Asp Asp Cys His Asp Ala Asp Leu
65 70 75 80
gtt gtc ctg acc gcg ggt gcg ccg cag aag cct ggc gaa acg cga ctg 288
Val Val Leu Thr Ala Gly Ala Pro Gln Lys Pro Gly Glu Thr Arg Leu
85 90 95
gat ctc gtc cat aag aac ttg aag atc aac aaa gaa atc gtc acc acc 336
Asp Leu Val His Lys Asn Leu Lys Ile Asn Lys Glu Ile Val Thr Thr
100 105 110
atc gtg gac tcg ggc ttc aac ggc atc ttc ctg gtg gcg gcc aac cca 384
Ile Val Asp Ser Gly Phe Asn Gly Ile Phe Leu Val Ala Ala Asn Pro
115 120 125
gta gac atc ctg acc tac tca acg tgg aag ttt agc ggg ttc ccg aag 432
Val Asp Ile Leu Thr Tyr Ser Thr Trp Lys Phe Ser Gly Phe Pro Lys
130 135 140
gaa cgg gtc atc ggg tcc ggc aca tcg ctt gac tcc gcg cgg ttc cgc 480
Glu Arg Val Ile Gly Ser Gly Thr Ser Leu Asp Ser Ala Arg Phe Arg
145 150 155 160
cag gcc att gcg gag ctg gtc gac gtg gac gcc cgc aac gtg cac gcg 528
Gln Ala Ile Ala Glu Leu Val Asp Val Asp Ala Arg Asn Val His Ala
165 170 175
tat atc ctc ggc gag cat ggc gac acc gag ttt ccc gtt tgg tcc cac 576
Tyr Ile Leu Gly Glu His Gly Asp Thr Glu Phe Pro Val Trp Ser His
180 185 190
gcc aat gtg gcg gga ttg cag atc tac gag tgg gta aag aat aat ccc 624
Ala Asn Val Ala Gly Leu Gln Ile Tyr Glu Trp Val Lys Asn Asn Pro
195 200 205
gat gtc gac gaa gaa gcc atg gtc aac ctc ttc ttc aac gtg cgc gat 672
Asp Val Asp Glu Glu Ala Met Val Asn Leu Phe Phe Asn Val Arg Asp
210 215 220
gca gct tat acg atc atc gag aaa aag gga gcc acg ttc tat ggc atc 720
Ala Ala Tyr Thr Ile Ile Glu Lys Lys Gly Ala Thr Phe Tyr Gly Ile
225 230 235 240
gcc gtg gcg ctg gcc cgg atc acg aag gcc atc ctg aac gac gag aat 768
Ala Val Ala Leu Ala Arg Ile Thr Lys Ala Ile Leu Asn Asp Glu Asn
245 250 255
tcc gtg ctc ccg ctg agt gtc tac ctg gaa ggg gag tac ggt cag aac 816
Ser Val Leu Pro Leu Ser Val Tyr Leu Glu Gly Glu Tyr Gly Gln Asn
260 265 270
gac atc tac atc ggc gca ccg gcc atc att aac cgt cag ggc gtc aaa 864
Asp Ile Tyr Ile Gly Ala Pro Ala Ile Ile Asn Arg Gln Gly Val Lys
275 280 285
caa gtg att gaa atc ccg ctg acc gac gcc gag caa gag aag atg gaa 912
Gln Val Ile Glu Ile Pro Leu Thr Asp Ala Glu Gln Glu Lys Met Glu
290 295 300
gcc agc gct tcg gcg ctg aaa gaa gtc atc gag act gcc ttc gcc aag 960
Ala Ser Ala Ser Ala Leu Lys Glu Val Ile Glu Thr Ala Phe Ala Lys
305 310 315 320
ttc gag gcc gaa gaa gcg aag taa 984
Phe Glu Ala Glu Glu Ala Lys
325
<210> 12
<211> 327
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 12
Met Thr Ala Ala Ala Gly Asn Lys Asp His Gln Lys Val Ile Leu Val
1 5 10 15
Gly Asp Gly Ala Val Gly Ser Ser Tyr Ala Phe Ala Leu Val Thr Gln
20 25 30
Asn Ile Ala Gln Glu Val Gly Ile Ile Asp Ile Asn Val Pro Lys Thr
35 40 45
Glu Gly Asp Ala Leu Asp Leu Ser His Ala Leu Ala Phe Thr Ser Pro
50 55 60
Lys Lys Ile Tyr Ala Ala Thr Tyr Asp Asp Cys His Asp Ala Asp Leu
65 70 75 80
Val Val Leu Thr Ala Gly Ala Pro Gln Lys Pro Gly Glu Thr Arg Leu
85 90 95
Asp Leu Val His Lys Asn Leu Lys Ile Asn Lys Glu Ile Val Thr Thr
100 105 110
Ile Val Asp Ser Gly Phe Asn Gly Ile Phe Leu Val Ala Ala Asn Pro
115 120 125
Val Asp Ile Leu Thr Tyr Ser Thr Trp Lys Phe Ser Gly Phe Pro Lys
130 135 140
Glu Arg Val Ile Gly Ser Gly Thr Ser Leu Asp Ser Ala Arg Phe Arg
145 150 155 160
Gln Ala Ile Ala Glu Leu Val Asp Val Asp Ala Arg Asn Val His Ala
165 170 175
Tyr Ile Leu Gly Glu His Gly Asp Thr Glu Phe Pro Val Trp Ser His
180 185 190
Ala Asn Val Ala Gly Leu Gln Ile Tyr Glu Trp Val Lys Asn Asn Pro
195 200 205
Asp Val Asp Glu Glu Ala Met Val Asn Leu Phe Phe Asn Val Arg Asp
210 215 220
Ala Ala Tyr Thr Ile Ile Glu Lys Lys Gly Ala Thr Phe Tyr Gly Ile
225 230 235 240
Ala Val Ala Leu Ala Arg Ile Thr Lys Ala Ile Leu Asn Asp Glu Asn
245 250 255
Ser Val Leu Pro Leu Ser Val Tyr Leu Glu Gly Glu Tyr Gly Gln Asn
260 265 270
Asp Ile Tyr Ile Gly Ala Pro Ala Ile Ile Asn Arg Gln Gly Val Lys
275 280 285
Gln Val Ile Glu Ile Pro Leu Thr Asp Ala Glu Gln Glu Lys Met Glu
290 295 300
Ala Ser Ala Ser Ala Leu Lys Glu Val Ile Glu Thr Ala Phe Ala Lys
305 310 315 320
Phe Glu Ala Glu Glu Ala Lys
325
<210> 13
<211> 939
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Lactobacillus casei)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(939)
<400> 13
atg cgc aat aac ggg aac atc att ctt ata ggt gac ggt gcc atc ggt 48
Met Arg Asn Asn Gly Asn Ile Ile Leu Ile Gly Asp Gly Ala Ile Gly
1 5 10 15
agt tct tat gcc ttc aac tgc ctg acc acc ggc gtc ggc cag tcc ctg 96
Ser Ser Tyr Ala Phe Asn Cys Leu Thr Thr Gly Val Gly Gln Ser Leu
20 25 30
ggg ata atc gac gtg aac gag aag cgc gta cag ggc gac gtc gag gac 144
Gly Ile Ile Asp Val Asn Glu Lys Arg Val Gln Gly Asp Val Glu Asp
35 40 45
ttg agc gac gcc ctg ccc tac act tcg cag aag aac atc tac gct gcg 192
Leu Ser Asp Ala Leu Pro Tyr Thr Ser Gln Lys Asn Ile Tyr Ala Ala
50 55 60
tcc tac gaa gat tgc aaa tat gcc gac att atc gtg atc acg gca ggc 240
Ser Tyr Glu Asp Cys Lys Tyr Ala Asp Ile Ile Val Ile Thr Ala Gly
65 70 75 80
atc gcc cag aag ccc gga cag acc cgg ttg cag ctc ctg gca atc aac 288
Ile Ala Gln Lys Pro Gly Gln Thr Arg Leu Gln Leu Leu Ala Ile Asn
85 90 95
gcg aag att atg aaa gaa atc acc cat aac atc atg gcc tcg ggt ttc 336
Ala Lys Ile Met Lys Glu Ile Thr His Asn Ile Met Ala Ser Gly Phe
100 105 110
aat ggc ttc atc ctg gtc gcc agc aac ccg gtg gac gtc ttg gcg gag 384
Asn Gly Phe Ile Leu Val Ala Ser Asn Pro Val Asp Val Leu Ala Glu
115 120 125
ctg gtg ctc cag gaa tcg ggc ctg ccc cgg aac caa gtc ctg ggc tcg 432
Leu Val Leu Gln Glu Ser Gly Leu Pro Arg Asn Gln Val Leu Gly Ser
130 135 140
ggc acc gcc ctc gac tcg gcg cgt ctg cgg agc gag atc ggg ctg cgc 480
Gly Thr Ala Leu Asp Ser Ala Arg Leu Arg Ser Glu Ile Gly Leu Arg
145 150 155 160
tac aat gtc gac gca cgg atc gtc cac ggg tac atc atg ggc gag cac 528
Tyr Asn Val Asp Ala Arg Ile Val His Gly Tyr Ile Met Gly Glu His
165 170 175
ggc gat tcc gag ttc ccg gtg tgg gac tac acc aat atc ggc ggc aag 576
Gly Asp Ser Glu Phe Pro Val Trp Asp Tyr Thr Asn Ile Gly Gly Lys
180 185 190
ccg atc ctc gat tgg atc ccg aag gac cgc cag gac aaa gat ctc cct 624
Pro Ile Leu Asp Trp Ile Pro Lys Asp Arg Gln Asp Lys Asp Leu Pro
195 200 205
gat atc tcc gaa agg gtt aag aca gcg gcc tat gga att atc gag aag 672
Asp Ile Ser Glu Arg Val Lys Thr Ala Ala Tyr Gly Ile Ile Glu Lys
210 215 220
aaa ggg gcc acg ttt tac ggc atc gcc gcg tcc ctg acc cgt ttg acg 720
Lys Gly Ala Thr Phe Tyr Gly Ile Ala Ala Ser Leu Thr Arg Leu Thr
225 230 235 240
tca gcg ttc ctg aac gac gat cga gcg gcc ttc gcc atg agc gtg cat 768
Ser Ala Phe Leu Asn Asp Asp Arg Ala Ala Phe Ala Met Ser Val His
245 250 255
ctg gaa ggc gaa tat ggc ctt agc gga gtg tcg atc ggc gtc ccg gtg 816
Leu Glu Gly Glu Tyr Gly Leu Ser Gly Val Ser Ile Gly Val Pro Val
260 265 270
atc ctg ggc gcc aac ggc ctg gag cgc atc atc gaa ctc gac ctg aat 864
Ile Leu Gly Ala Asn Gly Leu Glu Arg Ile Ile Glu Leu Asp Leu Asn
275 280 285
cca gag gat cac aag cgc ctg gcc gac agc gct gcg atc ctc aag gaa 912
Pro Glu Asp His Lys Arg Leu Ala Asp Ser Ala Ala Ile Leu Lys Glu
290 295 300
aac ctg aaa aag gcg caa gaa gcc taa 939
Asn Leu Lys Lys Ala Gln Glu Ala
305 310
<210> 14
<211> 312
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 14
Met Arg Asn Asn Gly Asn Ile Ile Leu Ile Gly Asp Gly Ala Ile Gly
1 5 10 15
Ser Ser Tyr Ala Phe Asn Cys Leu Thr Thr Gly Val Gly Gln Ser Leu
20 25 30
Gly Ile Ile Asp Val Asn Glu Lys Arg Val Gln Gly Asp Val Glu Asp
35 40 45
Leu Ser Asp Ala Leu Pro Tyr Thr Ser Gln Lys Asn Ile Tyr Ala Ala
50 55 60
Ser Tyr Glu Asp Cys Lys Tyr Ala Asp Ile Ile Val Ile Thr Ala Gly
65 70 75 80
Ile Ala Gln Lys Pro Gly Gln Thr Arg Leu Gln Leu Leu Ala Ile Asn
85 90 95
Ala Lys Ile Met Lys Glu Ile Thr His Asn Ile Met Ala Ser Gly Phe
100 105 110
Asn Gly Phe Ile Leu Val Ala Ser Asn Pro Val Asp Val Leu Ala Glu
115 120 125
Leu Val Leu Gln Glu Ser Gly Leu Pro Arg Asn Gln Val Leu Gly Ser
130 135 140
Gly Thr Ala Leu Asp Ser Ala Arg Leu Arg Ser Glu Ile Gly Leu Arg
145 150 155 160
Tyr Asn Val Asp Ala Arg Ile Val His Gly Tyr Ile Met Gly Glu His
165 170 175
Gly Asp Ser Glu Phe Pro Val Trp Asp Tyr Thr Asn Ile Gly Gly Lys
180 185 190
Pro Ile Leu Asp Trp Ile Pro Lys Asp Arg Gln Asp Lys Asp Leu Pro
195 200 205
Asp Ile Ser Glu Arg Val Lys Thr Ala Ala Tyr Gly Ile Ile Glu Lys
210 215 220
Lys Gly Ala Thr Phe Tyr Gly Ile Ala Ala Ser Leu Thr Arg Leu Thr
225 230 235 240
Ser Ala Phe Leu Asn Asp Asp Arg Ala Ala Phe Ala Met Ser Val His
245 250 255
Leu Glu Gly Glu Tyr Gly Leu Ser Gly Val Ser Ile Gly Val Pro Val
260 265 270
Ile Leu Gly Ala Asn Gly Leu Glu Arg Ile Ile Glu Leu Asp Leu Asn
275 280 285
Pro Glu Asp His Lys Arg Leu Ala Asp Ser Ala Ala Ile Leu Lys Glu
290 295 300
Asn Leu Lys Lys Ala Gln Glu Ala
305 310
<210> 15
<211> 957
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Bacillus megaterium)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(957)
<400> 15
atg aaa acg cag ttc acc ccc aag aca cga aaa gta gca gtt ata ggc 48
Met Lys Thr Gln Phe Thr Pro Lys Thr Arg Lys Val Ala Val Ile Gly
1 5 10 15
acc ggc ttc gtg ggc tcc tcc tac gcg ttc tcg atg gtc aat cag ggg 96
Thr Gly Phe Val Gly Ser Ser Tyr Ala Phe Ser Met Val Asn Gln Gly
20 25 30
atc gcg aac gaa ctg gtc ctg atc gac atg aac aag gaa aag gcc gag 144
Ile Ala Asn Glu Leu Val Leu Ile Asp Met Asn Lys Glu Lys Ala Glu
35 40 45
ggc gaa gcc cgg gat atc aac cat ggc atg cct ttt gcc acc ccg atg 192
Gly Glu Ala Arg Asp Ile Asn His Gly Met Pro Phe Ala Thr Pro Met
50 55 60
aag att tgg gcc gga gat tac aag gac tgc gcg gac gcg gac ctg gcc 240
Lys Ile Trp Ala Gly Asp Tyr Lys Asp Cys Ala Asp Ala Asp Leu Ala
65 70 75 80
gtg atc acc gcg ggt gcg aat cag gct ccg ggt gaa acc cgg ctg gac 288
Val Ile Thr Ala Gly Ala Asn Gln Ala Pro Gly Glu Thr Arg Leu Asp
85 90 95
ctc gtc gag aaa aat gtc aag atc ttc gag tgc atc gtt aaa gac atc 336
Leu Val Glu Lys Asn Val Lys Ile Phe Glu Cys Ile Val Lys Asp Ile
100 105 110
atg aac tcg ggg ttc gac ggc atc atc ctg gtc gcc acc aac ccg gtc 384
Met Asn Ser Gly Phe Asp Gly Ile Ile Leu Val Ala Thr Asn Pro Val
115 120 125
gat atc ctg gcc cat gtc acg caa aag gtg agc ggc ctg ccc aac gag 432
Asp Ile Leu Ala His Val Thr Gln Lys Val Ser Gly Leu Pro Asn Glu
130 135 140
cgg gtc att ggt tcg ggc acc atc ttg gac acg gcc cgt ttc cgc tat 480
Arg Val Ile Gly Ser Gly Thr Ile Leu Asp Thr Ala Arg Phe Arg Tyr
145 150 155 160
ctc ttg agc gat tac ttc gaa gtg gat tcc cgc aac gtc cac gcg tac 528
Leu Leu Ser Asp Tyr Phe Glu Val Asp Ser Arg Asn Val His Ala Tyr
165 170 175
atc atg ggc gag cac ggg gac acg gaa ttc ccg gtc tgg tcg cac gcc 576
Ile Met Gly Glu His Gly Asp Thr Glu Phe Pro Val Trp Ser His Ala
180 185 190
cag atc ggc gga gtg aag ctg gag cac ttt atc aat acg gca gct atc 624
Gln Ile Gly Gly Val Lys Leu Glu His Phe Ile Asn Thr Ala Ala Ile
195 200 205
gaa aaa gag cca gac atg cag cac ctg ttc gag cag acc cgg gac gcg 672
Glu Lys Glu Pro Asp Met Gln His Leu Phe Glu Gln Thr Arg Asp Ala
210 215 220
gcc tac cac atc att aat cgc aag ggc gcg acg tac tat gga atc gcc 720
Ala Tyr His Ile Ile Asn Arg Lys Gly Ala Thr Tyr Tyr Gly Ile Ala
225 230 235 240
atg ggg ctg gtc agg atc acc aag gcc atc ctt gac gat gag aac agc 768
Met Gly Leu Val Arg Ile Thr Lys Ala Ile Leu Asp Asp Glu Asn Ser
245 250 255
atc ctc acc gtg agt gcg ctg ttg gaa ggc cag tat ggc ata tca gac 816
Ile Leu Thr Val Ser Ala Leu Leu Glu Gly Gln Tyr Gly Ile Ser Asp
260 265 270
gtg tat atc ggt gtg ccc gcc att atc aac aag aac ggc gtg cgg cag 864
Val Tyr Ile Gly Val Pro Ala Ile Ile Asn Lys Asn Gly Val Arg Gln
275 280 285
atc atc gag ctg aac ctg acc ccg cat gaa caa cag cag ctc gaa cat 912
Ile Ile Glu Leu Asn Leu Thr Pro His Glu Gln Gln Gln Leu Glu His
290 295 300
agc gcc tcc atc ctc aag cag act cgc gat cgc gcc ttc gtg taa 957
Ser Ala Ser Ile Leu Lys Gln Thr Arg Asp Arg Ala Phe Val
305 310 315
<210> 16
<211> 318
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 16
Met Lys Thr Gln Phe Thr Pro Lys Thr Arg Lys Val Ala Val Ile Gly
1 5 10 15
Thr Gly Phe Val Gly Ser Ser Tyr Ala Phe Ser Met Val Asn Gln Gly
20 25 30
Ile Ala Asn Glu Leu Val Leu Ile Asp Met Asn Lys Glu Lys Ala Glu
35 40 45
Gly Glu Ala Arg Asp Ile Asn His Gly Met Pro Phe Ala Thr Pro Met
50 55 60
Lys Ile Trp Ala Gly Asp Tyr Lys Asp Cys Ala Asp Ala Asp Leu Ala
65 70 75 80
Val Ile Thr Ala Gly Ala Asn Gln Ala Pro Gly Glu Thr Arg Leu Asp
85 90 95
Leu Val Glu Lys Asn Val Lys Ile Phe Glu Cys Ile Val Lys Asp Ile
100 105 110
Met Asn Ser Gly Phe Asp Gly Ile Ile Leu Val Ala Thr Asn Pro Val
115 120 125
Asp Ile Leu Ala His Val Thr Gln Lys Val Ser Gly Leu Pro Asn Glu
130 135 140
Arg Val Ile Gly Ser Gly Thr Ile Leu Asp Thr Ala Arg Phe Arg Tyr
145 150 155 160
Leu Leu Ser Asp Tyr Phe Glu Val Asp Ser Arg Asn Val His Ala Tyr
165 170 175
Ile Met Gly Glu His Gly Asp Thr Glu Phe Pro Val Trp Ser His Ala
180 185 190
Gln Ile Gly Gly Val Lys Leu Glu His Phe Ile Asn Thr Ala Ala Ile
195 200 205
Glu Lys Glu Pro Asp Met Gln His Leu Phe Glu Gln Thr Arg Asp Ala
210 215 220
Ala Tyr His Ile Ile Asn Arg Lys Gly Ala Thr Tyr Tyr Gly Ile Ala
225 230 235 240
Met Gly Leu Val Arg Ile Thr Lys Ala Ile Leu Asp Asp Glu Asn Ser
245 250 255
Ile Leu Thr Val Ser Ala Leu Leu Glu Gly Gln Tyr Gly Ile Ser Asp
260 265 270
Val Tyr Ile Gly Val Pro Ala Ile Ile Asn Lys Asn Gly Val Arg Gln
275 280 285
Ile Ile Glu Leu Asn Leu Thr Pro His Glu Gln Gln Gln Leu Glu His
290 295 300
Ser Ala Ser Ile Leu Lys Gln Thr Arg Asp Arg Ala Phe Val
305 310 315
<210> 17
<211> 957
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Taeniopygia guttata)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(957)
<400> 17
atg aaa acg cag ttc acc ccc aag aca cga aaa gta gca gtt ata ggc 48
Met Lys Thr Gln Phe Thr Pro Lys Thr Arg Lys Val Ala Val Ile Gly
1 5 10 15
acc ggc ttc gtg ggc tcc tcc tac gcg ttc tcg atg gtc aat cag ggg 96
Thr Gly Phe Val Gly Ser Ser Tyr Ala Phe Ser Met Val Asn Gln Gly
20 25 30
atc gcg aac gaa ctg gtc ctg atc gac atg aac aag gaa aag gcc gag 144
Ile Ala Asn Glu Leu Val Leu Ile Asp Met Asn Lys Glu Lys Ala Glu
35 40 45
ggc gaa gcc cgg gat atc aac cat ggc atg cct ttt gcc acc ccg atg 192
Gly Glu Ala Arg Asp Ile Asn His Gly Met Pro Phe Ala Thr Pro Met
50 55 60
aag att tgg gcc gga gat tac aag gac tgc gcg gac gcg gac ctg gcc 240
Lys Ile Trp Ala Gly Asp Tyr Lys Asp Cys Ala Asp Ala Asp Leu Ala
65 70 75 80
gtg atc acc gcg ggt gcg aat cag gct ccg ggt gaa acc cgg ctg gac 288
Val Ile Thr Ala Gly Ala Asn Gln Ala Pro Gly Glu Thr Arg Leu Asp
85 90 95
ctc gtc gag aaa aat gtc aag atc ttc gag tgc atc gtt aaa gac atc 336
Leu Val Glu Lys Asn Val Lys Ile Phe Glu Cys Ile Val Lys Asp Ile
100 105 110
atg aac tcg ggg ttc gac ggc atc atc ctg gtc gcc acc aac ccg gtc 384
Met Asn Ser Gly Phe Asp Gly Ile Ile Leu Val Ala Thr Asn Pro Val
115 120 125
gat atc ctg gcc cat gtc acg caa aag gtg agc ggc ctg ccc aac gag 432
Asp Ile Leu Ala His Val Thr Gln Lys Val Ser Gly Leu Pro Asn Glu
130 135 140
cgg gtc att ggt tcg ggc acc atc ttg gac acg gcc cgt ttc cgc tat 480
Arg Val Ile Gly Ser Gly Thr Ile Leu Asp Thr Ala Arg Phe Arg Tyr
145 150 155 160
ctc ttg agc gat tac ttc gaa gtg gat tcc cgc aac gtc cac gcg tac 528
Leu Leu Ser Asp Tyr Phe Glu Val Asp Ser Arg Asn Val His Ala Tyr
165 170 175
atc atg ggc gag cac ggg gac acg gaa ttc ccg gtc tgg tcg cac gcc 576
Ile Met Gly Glu His Gly Asp Thr Glu Phe Pro Val Trp Ser His Ala
180 185 190
cag atc ggc gga gtg aag ctg gag cac ttt atc aat acg gca gct atc 624
Gln Ile Gly Gly Val Lys Leu Glu His Phe Ile Asn Thr Ala Ala Ile
195 200 205
gaa aaa gag cca gac atg cag cac ctg ttc gag cag acc cgg gac gcg 672
Glu Lys Glu Pro Asp Met Gln His Leu Phe Glu Gln Thr Arg Asp Ala
210 215 220
gcc tac cac atc att aat cgc aag ggc gcg acg tac tat gga atc gcc 720
Ala Tyr His Ile Ile Asn Arg Lys Gly Ala Thr Tyr Tyr Gly Ile Ala
225 230 235 240
atg ggg ctg gtc agg atc acc aag gcc atc ctt gac gat gag aac agc 768
Met Gly Leu Val Arg Ile Thr Lys Ala Ile Leu Asp Asp Glu Asn Ser
245 250 255
atc ctc acc gtg agt gcg ctg ttg gaa ggc cag tat ggc ata tca gac 816
Ile Leu Thr Val Ser Ala Leu Leu Glu Gly Gln Tyr Gly Ile Ser Asp
260 265 270
gtg tat atc ggt gtg ccc gcc att atc aac aag aac ggc gtg cgg cag 864
Val Tyr Ile Gly Val Pro Ala Ile Ile Asn Lys Asn Gly Val Arg Gln
275 280 285
atc atc gag ctg aac ctg acc ccg cat gaa caa cag cag ctc gaa cat 912
Ile Ile Glu Leu Asn Leu Thr Pro His Glu Gln Gln Gln Leu Glu His
290 295 300
agc gcc tcc atc ctc aag cag act cgc gat cgc gcc ttc gtg taa 957
Ser Ala Ser Ile Leu Lys Gln Thr Arg Asp Arg Ala Phe Val
305 310 315
<210> 18
<211> 318
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 18
Met Lys Thr Gln Phe Thr Pro Lys Thr Arg Lys Val Ala Val Ile Gly
1 5 10 15
Thr Gly Phe Val Gly Ser Ser Tyr Ala Phe Ser Met Val Asn Gln Gly
20 25 30
Ile Ala Asn Glu Leu Val Leu Ile Asp Met Asn Lys Glu Lys Ala Glu
35 40 45
Gly Glu Ala Arg Asp Ile Asn His Gly Met Pro Phe Ala Thr Pro Met
50 55 60
Lys Ile Trp Ala Gly Asp Tyr Lys Asp Cys Ala Asp Ala Asp Leu Ala
65 70 75 80
Val Ile Thr Ala Gly Ala Asn Gln Ala Pro Gly Glu Thr Arg Leu Asp
85 90 95
Leu Val Glu Lys Asn Val Lys Ile Phe Glu Cys Ile Val Lys Asp Ile
100 105 110
Met Asn Ser Gly Phe Asp Gly Ile Ile Leu Val Ala Thr Asn Pro Val
115 120 125
Asp Ile Leu Ala His Val Thr Gln Lys Val Ser Gly Leu Pro Asn Glu
130 135 140
Arg Val Ile Gly Ser Gly Thr Ile Leu Asp Thr Ala Arg Phe Arg Tyr
145 150 155 160
Leu Leu Ser Asp Tyr Phe Glu Val Asp Ser Arg Asn Val His Ala Tyr
165 170 175
Ile Met Gly Glu His Gly Asp Thr Glu Phe Pro Val Trp Ser His Ala
180 185 190
Gln Ile Gly Gly Val Lys Leu Glu His Phe Ile Asn Thr Ala Ala Ile
195 200 205
Glu Lys Glu Pro Asp Met Gln His Leu Phe Glu Gln Thr Arg Asp Ala
210 215 220
Ala Tyr His Ile Ile Asn Arg Lys Gly Ala Thr Tyr Tyr Gly Ile Ala
225 230 235 240
Met Gly Leu Val Arg Ile Thr Lys Ala Ile Leu Asp Asp Glu Asn Ser
245 250 255
Ile Leu Thr Val Ser Ala Leu Leu Glu Gly Gln Tyr Gly Ile Ser Asp
260 265 270
Val Tyr Ile Gly Val Pro Ala Ile Ile Asn Lys Asn Gly Val Arg Gln
275 280 285
Ile Ile Glu Leu Asn Leu Thr Pro His Glu Gln Gln Gln Leu Glu His
290 295 300
Ser Ala Ser Ile Leu Lys Gln Thr Arg Asp Arg Ala Phe Val
305 310 315
<210> 19
<211> 963
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Lactobacillus plantarum)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(963)
<400> 19
atg tcg tcg atg cct aac cat cag aaa gtc gtc ctg gtg ggc gac gga 48
Met Ser Ser Met Pro Asn His Gln Lys Val Val Leu Val Gly Asp Gly
1 5 10 15
gcg gtc ggc agc tca tac gcc ttc gcg atg gcg caa cag gga atc gcg 96
Ala Val Gly Ser Ser Tyr Ala Phe Ala Met Ala Gln Gln Gly Ile Ala
20 25 30
gaa gaa ttc gtc atc gtc gac gtg gtg aag gac cgc acc aag ggc gac 144
Glu Glu Phe Val Ile Val Asp Val Val Lys Asp Arg Thr Lys Gly Asp
35 40 45
gcc ttg gac ctc gaa gat gca cag gcc ttc acg gcc ccg aag aaa atc 192
Ala Leu Asp Leu Glu Asp Ala Gln Ala Phe Thr Ala Pro Lys Lys Ile
50 55 60
tac agc ggc gag tat agc gac tgc aaa gac gcg gac ctc gtg gtc ata 240
Tyr Ser Gly Glu Tyr Ser Asp Cys Lys Asp Ala Asp Leu Val Val Ile
65 70 75 80
acc gcg gga gcg cca cag aag ccc gga gaa tcg agg ctg gac ctt gtc 288
Thr Ala Gly Ala Pro Gln Lys Pro Gly Glu Ser Arg Leu Asp Leu Val
85 90 95
aac aaa aac ttg aac atc ctg tcc tcg atc gtg aag ccc gtg gta gac 336
Asn Lys Asn Leu Asn Ile Leu Ser Ser Ile Val Lys Pro Val Val Asp
100 105 110
tcc ggg ttc gac ggc atc ttc ctg gtg gcc gcc aac ccc gtc gac atc 384
Ser Gly Phe Asp Gly Ile Phe Leu Val Ala Ala Asn Pro Val Asp Ile
115 120 125
ctc acc tat gcg acc tgg aag ttc agc ggg ttt ccg aag gac cgg gtc 432
Leu Thr Tyr Ala Thr Trp Lys Phe Ser Gly Phe Pro Lys Asp Arg Val
130 135 140
att ggc agt ggg acg tcg ctg gat agc tcg cgt ctg cga gtt gct ctg 480
Ile Gly Ser Gly Thr Ser Leu Asp Ser Ser Arg Leu Arg Val Ala Leu
145 150 155 160
ggg aag caa ttc aac gtg gat ccg cgc tct gtg gat gcg tat atc atg 528
Gly Lys Gln Phe Asn Val Asp Pro Arg Ser Val Asp Ala Tyr Ile Met
165 170 175
ggc gag cac ggc gat agt gag ttc gcc gca tat tcc acc gcc acg atc 576
Gly Glu His Gly Asp Ser Glu Phe Ala Ala Tyr Ser Thr Ala Thr Ile
180 185 190
ggc act cgg ccc gtg cgc gac gtt gcg aag gaa cag ggc gta tcc gac 624
Gly Thr Arg Pro Val Arg Asp Val Ala Lys Glu Gln Gly Val Ser Asp
195 200 205
gag gac ctg gcc aag ctg gaa gat ggc gtc cgc aac aag gcc tac gac 672
Glu Asp Leu Ala Lys Leu Glu Asp Gly Val Arg Asn Lys Ala Tyr Asp
210 215 220
atc atc aat ctc aag ggc gcg acc ttc tac ggt att ggt acc gcc ctg 720
Ile Ile Asn Leu Lys Gly Ala Thr Phe Tyr Gly Ile Gly Thr Ala Leu
225 230 235 240
atg cgg atc tcc aag gcc atc ctg cgc gat gag aac gcc gtg ttg ccg 768
Met Arg Ile Ser Lys Ala Ile Leu Arg Asp Glu Asn Ala Val Leu Pro
245 250 255
gtg ggg gct tac atg gat ggc cag tac ggc ctg aac gat atc tac atc 816
Val Gly Ala Tyr Met Asp Gly Gln Tyr Gly Leu Asn Asp Ile Tyr Ile
260 265 270
ggc aca ccg gcc gtc atc ggt ggc acc ggt ctg aag cag atc atc gag 864
Gly Thr Pro Ala Val Ile Gly Gly Thr Gly Leu Lys Gln Ile Ile Glu
275 280 285
agc ccg ctg tcc gcc gac gag ctg aaa aag atg cag gac agc gcc gca 912
Ser Pro Leu Ser Ala Asp Glu Leu Lys Lys Met Gln Asp Ser Ala Ala
290 295 300
acg ctc aaa aaa gtc ctc aat gat ggc ctg gcc gaa ctg gag aat aag 960
Thr Leu Lys Lys Val Leu Asn Asp Gly Leu Ala Glu Leu Glu Asn Lys
305 310 315 320
taa 963
<210> 20
<211> 320
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 20
Met Ser Ser Met Pro Asn His Gln Lys Val Val Leu Val Gly Asp Gly
1 5 10 15
Ala Val Gly Ser Ser Tyr Ala Phe Ala Met Ala Gln Gln Gly Ile Ala
20 25 30
Glu Glu Phe Val Ile Val Asp Val Val Lys Asp Arg Thr Lys Gly Asp
35 40 45
Ala Leu Asp Leu Glu Asp Ala Gln Ala Phe Thr Ala Pro Lys Lys Ile
50 55 60
Tyr Ser Gly Glu Tyr Ser Asp Cys Lys Asp Ala Asp Leu Val Val Ile
65 70 75 80
Thr Ala Gly Ala Pro Gln Lys Pro Gly Glu Ser Arg Leu Asp Leu Val
85 90 95
Asn Lys Asn Leu Asn Ile Leu Ser Ser Ile Val Lys Pro Val Val Asp
100 105 110
Ser Gly Phe Asp Gly Ile Phe Leu Val Ala Ala Asn Pro Val Asp Ile
115 120 125
Leu Thr Tyr Ala Thr Trp Lys Phe Ser Gly Phe Pro Lys Asp Arg Val
130 135 140
Ile Gly Ser Gly Thr Ser Leu Asp Ser Ser Arg Leu Arg Val Ala Leu
145 150 155 160
Gly Lys Gln Phe Asn Val Asp Pro Arg Ser Val Asp Ala Tyr Ile Met
165 170 175
Gly Glu His Gly Asp Ser Glu Phe Ala Ala Tyr Ser Thr Ala Thr Ile
180 185 190
Gly Thr Arg Pro Val Arg Asp Val Ala Lys Glu Gln Gly Val Ser Asp
195 200 205
Glu Asp Leu Ala Lys Leu Glu Asp Gly Val Arg Asn Lys Ala Tyr Asp
210 215 220
Ile Ile Asn Leu Lys Gly Ala Thr Phe Tyr Gly Ile Gly Thr Ala Leu
225 230 235 240
Met Arg Ile Ser Lys Ala Ile Leu Arg Asp Glu Asn Ala Val Leu Pro
245 250 255
Val Gly Ala Tyr Met Asp Gly Gln Tyr Gly Leu Asn Asp Ile Tyr Ile
260 265 270
Gly Thr Pro Ala Val Ile Gly Gly Thr Gly Leu Lys Gln Ile Ile Glu
275 280 285
Ser Pro Leu Ser Ala Asp Glu Leu Lys Lys Met Gln Asp Ser Ala Ala
290 295 300
Thr Leu Lys Lys Val Leu Asn Asp Gly Leu Ala Glu Leu Glu Asn Lys
305 310 315 320
<210> 21
<211> 963
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Lactobacillus acidophilus)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(963)
<400> 21
atg tcg tcg atg cct aac cat cag aaa gtc gtc ctg gtg ggc gac gga 48
Met Ser Ser Met Pro Asn His Gln Lys Val Val Leu Val Gly Asp Gly
1 5 10 15
gcg gtc ggc agc tca tac gcc ttc gcg atg gcg caa cag gga atc gcg 96
Ala Val Gly Ser Ser Tyr Ala Phe Ala Met Ala Gln Gln Gly Ile Ala
20 25 30
gaa gaa ttc gtc atc gtc gac gtg gtg aag gac cgc acc aag ggc gac 144
Glu Glu Phe Val Ile Val Asp Val Val Lys Asp Arg Thr Lys Gly Asp
35 40 45
gcc ttg gac ctc gaa gat gca cag gcc ttc acg gcc ccg aag aaa atc 192
Ala Leu Asp Leu Glu Asp Ala Gln Ala Phe Thr Ala Pro Lys Lys Ile
50 55 60
tac agc ggc gag tat agc gac tgc aaa gac gcg gac ctc gtg gtc ata 240
Tyr Ser Gly Glu Tyr Ser Asp Cys Lys Asp Ala Asp Leu Val Val Ile
65 70 75 80
acc gcg gga gcg cca cag aag ccc gga gaa tcg agg ctg gac ctt gtc 288
Thr Ala Gly Ala Pro Gln Lys Pro Gly Glu Ser Arg Leu Asp Leu Val
85 90 95
aac aaa aac ttg aac atc ctg tcc tcg atc gtg aag ccc gtg gta gac 336
Asn Lys Asn Leu Asn Ile Leu Ser Ser Ile Val Lys Pro Val Val Asp
100 105 110
tcc ggg ttc gac ggc atc ttc ctg gtg gcc gcc aac ccc gtc gac atc 384
Ser Gly Phe Asp Gly Ile Phe Leu Val Ala Ala Asn Pro Val Asp Ile
115 120 125
ctc acc tat gcg acc tgg aag ttc agc ggg ttt ccg aag gac cgg gtc 432
Leu Thr Tyr Ala Thr Trp Lys Phe Ser Gly Phe Pro Lys Asp Arg Val
130 135 140
att ggc agt ggg acg tcg ctg gat agc tcg cgt ctg cga gtt gct ctg 480
Ile Gly Ser Gly Thr Ser Leu Asp Ser Ser Arg Leu Arg Val Ala Leu
145 150 155 160
ggg aag caa ttc aac gtg gat ccg cgc tct gtg gat gcg tat atc atg 528
Gly Lys Gln Phe Asn Val Asp Pro Arg Ser Val Asp Ala Tyr Ile Met
165 170 175
ggc gag cac ggc gat agt gag ttc gcc gca tat tcc acc gcc acg atc 576
Gly Glu His Gly Asp Ser Glu Phe Ala Ala Tyr Ser Thr Ala Thr Ile
180 185 190
ggc act cgg ccc gtg cgc gac gtt gcg aag gaa cag ggc gta tcc gac 624
Gly Thr Arg Pro Val Arg Asp Val Ala Lys Glu Gln Gly Val Ser Asp
195 200 205
gag gac ctg gcc aag ctg gaa gat ggc gtc cgc aac aag gcc tac gac 672
Glu Asp Leu Ala Lys Leu Glu Asp Gly Val Arg Asn Lys Ala Tyr Asp
210 215 220
atc atc aat ctc aag ggc gcg acc ttc tac ggt att ggt acc gcc ctg 720
Ile Ile Asn Leu Lys Gly Ala Thr Phe Tyr Gly Ile Gly Thr Ala Leu
225 230 235 240
atg cgg atc tcc aag gcc atc ctg cgc gat gag aac gcc gtg ttg ccg 768
Met Arg Ile Ser Lys Ala Ile Leu Arg Asp Glu Asn Ala Val Leu Pro
245 250 255
gtg ggg gct tac atg gat ggc cag tac ggc ctg aac gat atc tac atc 816
Val Gly Ala Tyr Met Asp Gly Gln Tyr Gly Leu Asn Asp Ile Tyr Ile
260 265 270
ggc aca ccg gcc gtc atc ggt ggc acc ggt ctg aag cag atc atc gag 864
Gly Thr Pro Ala Val Ile Gly Gly Thr Gly Leu Lys Gln Ile Ile Glu
275 280 285
agc ccg ctg tcc gcc gac gag ctg aaa aag atg cag gac agc gcc gca 912
Ser Pro Leu Ser Ala Asp Glu Leu Lys Lys Met Gln Asp Ser Ala Ala
290 295 300
acg ctc aaa aaa gtc ctc aat gat ggc ctg gcc gaa ctg gag aat aag 960
Thr Leu Lys Lys Val Leu Asn Asp Gly Leu Ala Glu Leu Glu Asn Lys
305 310 315 320
taa 963
<210> 22
<211> 320
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 22
Met Ser Ser Met Pro Asn His Gln Lys Val Val Leu Val Gly Asp Gly
1 5 10 15
Ala Val Gly Ser Ser Tyr Ala Phe Ala Met Ala Gln Gln Gly Ile Ala
20 25 30
Glu Glu Phe Val Ile Val Asp Val Val Lys Asp Arg Thr Lys Gly Asp
35 40 45
Ala Leu Asp Leu Glu Asp Ala Gln Ala Phe Thr Ala Pro Lys Lys Ile
50 55 60
Tyr Ser Gly Glu Tyr Ser Asp Cys Lys Asp Ala Asp Leu Val Val Ile
65 70 75 80
Thr Ala Gly Ala Pro Gln Lys Pro Gly Glu Ser Arg Leu Asp Leu Val
85 90 95
Asn Lys Asn Leu Asn Ile Leu Ser Ser Ile Val Lys Pro Val Val Asp
100 105 110
Ser Gly Phe Asp Gly Ile Phe Leu Val Ala Ala Asn Pro Val Asp Ile
115 120 125
Leu Thr Tyr Ala Thr Trp Lys Phe Ser Gly Phe Pro Lys Asp Arg Val
130 135 140
Ile Gly Ser Gly Thr Ser Leu Asp Ser Ser Arg Leu Arg Val Ala Leu
145 150 155 160
Gly Lys Gln Phe Asn Val Asp Pro Arg Ser Val Asp Ala Tyr Ile Met
165 170 175
Gly Glu His Gly Asp Ser Glu Phe Ala Ala Tyr Ser Thr Ala Thr Ile
180 185 190
Gly Thr Arg Pro Val Arg Asp Val Ala Lys Glu Gln Gly Val Ser Asp
195 200 205
Glu Asp Leu Ala Lys Leu Glu Asp Gly Val Arg Asn Lys Ala Tyr Asp
210 215 220
Ile Ile Asn Leu Lys Gly Ala Thr Phe Tyr Gly Ile Gly Thr Ala Leu
225 230 235 240
Met Arg Ile Ser Lys Ala Ile Leu Arg Asp Glu Asn Ala Val Leu Pro
245 250 255
Val Gly Ala Tyr Met Asp Gly Gln Tyr Gly Leu Asn Asp Ile Tyr Ile
260 265 270
Gly Thr Pro Ala Val Ile Gly Gly Thr Gly Leu Lys Gln Ile Ile Glu
275 280 285
Ser Pro Leu Ser Ala Asp Glu Leu Lys Lys Met Gln Asp Ser Ala Ala
290 295 300
Thr Leu Lys Lys Val Leu Asn Asp Gly Leu Ala Glu Leu Glu Asn Lys
305 310 315 320
<210> 23
<211> 954
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Staphylococcus aureus)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(954)
<400> 23
atg aac aag ttc aag ggc aac aaa gtc gtg ttg atc ggg aat ggc gcc 48
Met Asn Lys Phe Lys Gly Asn Lys Val Val Leu Ile Gly Asn Gly Ala
1 5 10 15
gtc ggc tcg tcc tac gcg ttt tcg ctg gtc aac cag tcc atc gtg gac 96
Val Gly Ser Ser Tyr Ala Phe Ser Leu Val Asn Gln Ser Ile Val Asp
20 25 30
gaa ctc gtc ata atc gac ctc gac acc gaa aag gtt cgg ggt gac gtc 144
Glu Leu Val Ile Ile Asp Leu Asp Thr Glu Lys Val Arg Gly Asp Val
35 40 45
atg gac ctc aag cac gcc act ccg tat agc ccc acg acc gtg cgc gtg 192
Met Asp Leu Lys His Ala Thr Pro Tyr Ser Pro Thr Thr Val Arg Val
50 55 60
aag gcc gga gag tac agc gac tgc cat gac gcc aac ctg gtc gtc att 240
Lys Ala Gly Glu Tyr Ser Asp Cys His Asp Ala Asn Leu Val Val Ile
65 70 75 80
tgc gcg ggt gcg gcg cag aaa ccg ggc gaa acc cgc ctc gat ctg gtg 288
Cys Ala Gly Ala Ala Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val
85 90 95
agc aag aac ctc aag atc ttc aag agt att gtg gga gaa gta atg gcc 336
Ser Lys Asn Leu Lys Ile Phe Lys Ser Ile Val Gly Glu Val Met Ala
100 105 110
tcg aag ttc gac ggc atc ttc ctg gtc gca acc aat cca gtg gat atc 384
Ser Lys Phe Asp Gly Ile Phe Leu Val Ala Thr Asn Pro Val Asp Ile
115 120 125
ctg gcc tac gca acc tgg aag ttc tcg gga ctg ccc aaa gag cga gtc 432
Leu Ala Tyr Ala Thr Trp Lys Phe Ser Gly Leu Pro Lys Glu Arg Val
130 135 140
atc ggc tcc ggc acc atc ctt gac tca gcg cgt ttc cgg ctg ctg ttg 480
Ile Gly Ser Gly Thr Ile Leu Asp Ser Ala Arg Phe Arg Leu Leu Leu
145 150 155 160
agc gag gcc ttc gat gtg gcg ccc agg tct gtc gat gcc cag atc atc 528
Ser Glu Ala Phe Asp Val Ala Pro Arg Ser Val Asp Ala Gln Ile Ile
165 170 175
ggc gag cac ggc gac acc gag ctt ccc gtg tgg tcc cat gcc aat atc 576
Gly Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser His Ala Asn Ile
180 185 190
gct ggg cag ccg ctg aaa acg ctg ctg gag caa cgt cct gag ggc aag 624
Ala Gly Gln Pro Leu Lys Thr Leu Leu Glu Gln Arg Pro Glu Gly Lys
195 200 205
gcg cag atc gag cag atc ttt gtg cag acg cgc gac gcc gcc tac gac 672
Ala Gln Ile Glu Gln Ile Phe Val Gln Thr Arg Asp Ala Ala Tyr Asp
210 215 220
atc atc caa gcg aag ggc gct aca tac tat ggg gtg gcg atg ggg ctc 720
Ile Ile Gln Ala Lys Gly Ala Thr Tyr Tyr Gly Val Ala Met Gly Leu
225 230 235 240
gcg cgc atc acg gaa gcc att ttc cgg aat gaa gat gca gtc ctg acc 768
Ala Arg Ile Thr Glu Ala Ile Phe Arg Asn Glu Asp Ala Val Leu Thr
245 250 255
gta agc gcc ctg ctg gaa ggt gag tat gac gaa gaa gat gtc tac atc 816
Val Ser Ala Leu Leu Glu Gly Glu Tyr Asp Glu Glu Asp Val Tyr Ile
260 265 270
ggc gtt ccg gcc gtg atc aac cgc aac ggc atc cgg aac gtc gtg gag 864
Gly Val Pro Ala Val Ile Asn Arg Asn Gly Ile Arg Asn Val Val Glu
275 280 285
atc ccg ctg aac gat gaa gaa cag agc aag ttc gcc cac tcc gcg aaa 912
Ile Pro Leu Asn Asp Glu Glu Gln Ser Lys Phe Ala His Ser Ala Lys
290 295 300
acc ctg aaa gac atc atg gcc gag gcc gag gaa ttg aag taa 954
Thr Leu Lys Asp Ile Met Ala Glu Ala Glu Glu Leu Lys
305 310 315
<210> 24
<211> 317
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 24
Met Asn Lys Phe Lys Gly Asn Lys Val Val Leu Ile Gly Asn Gly Ala
1 5 10 15
Val Gly Ser Ser Tyr Ala Phe Ser Leu Val Asn Gln Ser Ile Val Asp
20 25 30
Glu Leu Val Ile Ile Asp Leu Asp Thr Glu Lys Val Arg Gly Asp Val
35 40 45
Met Asp Leu Lys His Ala Thr Pro Tyr Ser Pro Thr Thr Val Arg Val
50 55 60
Lys Ala Gly Glu Tyr Ser Asp Cys His Asp Ala Asn Leu Val Val Ile
65 70 75 80
Cys Ala Gly Ala Ala Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val
85 90 95
Ser Lys Asn Leu Lys Ile Phe Lys Ser Ile Val Gly Glu Val Met Ala
100 105 110
Ser Lys Phe Asp Gly Ile Phe Leu Val Ala Thr Asn Pro Val Asp Ile
115 120 125
Leu Ala Tyr Ala Thr Trp Lys Phe Ser Gly Leu Pro Lys Glu Arg Val
130 135 140
Ile Gly Ser Gly Thr Ile Leu Asp Ser Ala Arg Phe Arg Leu Leu Leu
145 150 155 160
Ser Glu Ala Phe Asp Val Ala Pro Arg Ser Val Asp Ala Gln Ile Ile
165 170 175
Gly Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser His Ala Asn Ile
180 185 190
Ala Gly Gln Pro Leu Lys Thr Leu Leu Glu Gln Arg Pro Glu Gly Lys
195 200 205
Ala Gln Ile Glu Gln Ile Phe Val Gln Thr Arg Asp Ala Ala Tyr Asp
210 215 220
Ile Ile Gln Ala Lys Gly Ala Thr Tyr Tyr Gly Val Ala Met Gly Leu
225 230 235 240
Ala Arg Ile Thr Glu Ala Ile Phe Arg Asn Glu Asp Ala Val Leu Thr
245 250 255
Val Ser Ala Leu Leu Glu Gly Glu Tyr Asp Glu Glu Asp Val Tyr Ile
260 265 270
Gly Val Pro Ala Val Ile Asn Arg Asn Gly Ile Arg Asn Val Val Glu
275 280 285
Ile Pro Leu Asn Asp Glu Glu Gln Ser Lys Phe Ala His Ser Ala Lys
290 295 300
Thr Leu Lys Asp Ile Met Ala Glu Ala Glu Glu Leu Lys
305 310 315
<210> 25
<211> 954
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Bacillus caldolyticus)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(954)
<400> 25
atg aaa aac aat ggg ggc acc cgc gtg gtc gtc atc gga acc ggc ttc 48
Met Lys Asn Asn Gly Gly Thr Arg Val Val Val Ile Gly Thr Gly Phe
1 5 10 15
gtc ggc gcc tcg tac gcg ttc gcc ctg atg aac cag ggt att gcc gac 96
Val Gly Ala Ser Tyr Ala Phe Ala Leu Met Asn Gln Gly Ile Ala Asp
20 25 30
gag atc gtg ctg atc gac gcg aac gag tcc aag gcc atc ggc gat gca 144
Glu Ile Val Leu Ile Asp Ala Asn Glu Ser Lys Ala Ile Gly Asp Ala
35 40 45
atg gac ttt aac cac ggc aag gtt ttc gcc ccc aaa ccg gcg gac atc 192
Met Asp Phe Asn His Gly Lys Val Phe Ala Pro Lys Pro Ala Asp Ile
50 55 60
tgg cat ggc gat tat gac gac tgc cgg gac gcc gac ctg gtg gtg atc 240
Trp His Gly Asp Tyr Asp Asp Cys Arg Asp Ala Asp Leu Val Val Ile
65 70 75 80
tgc gcg ggt gcg aac cag aag ccg gga gaa acc cgc ctt gac ctg gtt 288
Cys Ala Gly Ala Asn Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val
85 90 95
gac aag aac atc gcc atc ttc cgc tcg atc gtc gaa agc gtc atg gcc 336
Asp Lys Asn Ile Ala Ile Phe Arg Ser Ile Val Glu Ser Val Met Ala
100 105 110
agc ggg ttc caa ggc ctc ttc ctg gtc gcc acc aac ccc gtc gac att 384
Ser Gly Phe Gln Gly Leu Phe Leu Val Ala Thr Asn Pro Val Asp Ile
115 120 125
ctc acg tac gcg acc tgg aaa ttc tcg ggc ctg ccg cac gag cgg gtg 432
Leu Thr Tyr Ala Thr Trp Lys Phe Ser Gly Leu Pro His Glu Arg Val
130 135 140
atc ggc agc ggc acg atc ctg gat act gcc cgg ttt cga ttc ttg ctg 480
Ile Gly Ser Gly Thr Ile Leu Asp Thr Ala Arg Phe Arg Phe Leu Leu
145 150 155 160
ggt gag tac ttc agc gtc gcc cca cag aat gtg cat gcg tac atc att 528
Gly Glu Tyr Phe Ser Val Ala Pro Gln Asn Val His Ala Tyr Ile Ile
165 170 175
ggc gaa cat ggc gat acg gaa ctc ccc gtc tgg tcc cag gct gat atc 576
Gly Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser Gln Ala Asp Ile
180 185 190
ggg gga gtc ccg atc cgg aag ctc gtg gaa tcc aag ggc gaa gaa gcg 624
Gly Gly Val Pro Ile Arg Lys Leu Val Glu Ser Lys Gly Glu Glu Ala
195 200 205
cag aaa gaa ttg gag cgt atc ttc gtg aat gta cgt gat gcc gcc tat 672
Gln Lys Glu Leu Glu Arg Ile Phe Val Asn Val Arg Asp Ala Ala Tyr
210 215 220
cag ata atc gaa aag aaa ggc gcc acc tac tat ggt atc gcg atg ggg 720
Gln Ile Ile Glu Lys Lys Gly Ala Thr Tyr Tyr Gly Ile Ala Met Gly
225 230 235 240
ctg gcc agg gtg acc cgc gca atc ctg cac aac gag aat gcg atc ctg 768
Leu Ala Arg Val Thr Arg Ala Ile Leu His Asn Glu Asn Ala Ile Leu
245 250 255
aca gtg tcc gcg tac ctg gat ggg cct tac ggc gag cgc gac gtg tat 816
Thr Val Ser Ala Tyr Leu Asp Gly Pro Tyr Gly Glu Arg Asp Val Tyr
260 265 270
atc ggc gtg ccg gca gtc atc aac cgg aac ggc atc cgc gaa gtc atc 864
Ile Gly Val Pro Ala Val Ile Asn Arg Asn Gly Ile Arg Glu Val Ile
275 280 285
gag atc gag ctg aac gac gac gag aag aac cgc ttc cac cac tcg gcc 912
Glu Ile Glu Leu Asn Asp Asp Glu Lys Asn Arg Phe His His Ser Ala
290 295 300
gct acc ctc aag agt gtg ttg gcc cgg ttc ttc acg cgc taa 954
Ala Thr Leu Lys Ser Val Leu Ala Arg Phe Phe Thr Arg
305 310 315
<210> 26
<211> 317
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 26
Met Lys Asn Asn Gly Gly Thr Arg Val Val Val Ile Gly Thr Gly Phe
1 5 10 15
Val Gly Ala Ser Tyr Ala Phe Ala Leu Met Asn Gln Gly Ile Ala Asp
20 25 30
Glu Ile Val Leu Ile Asp Ala Asn Glu Ser Lys Ala Ile Gly Asp Ala
35 40 45
Met Asp Phe Asn His Gly Lys Val Phe Ala Pro Lys Pro Ala Asp Ile
50 55 60
Trp His Gly Asp Tyr Asp Asp Cys Arg Asp Ala Asp Leu Val Val Ile
65 70 75 80
Cys Ala Gly Ala Asn Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val
85 90 95
Asp Lys Asn Ile Ala Ile Phe Arg Ser Ile Val Glu Ser Val Met Ala
100 105 110
Ser Gly Phe Gln Gly Leu Phe Leu Val Ala Thr Asn Pro Val Asp Ile
115 120 125
Leu Thr Tyr Ala Thr Trp Lys Phe Ser Gly Leu Pro His Glu Arg Val
130 135 140
Ile Gly Ser Gly Thr Ile Leu Asp Thr Ala Arg Phe Arg Phe Leu Leu
145 150 155 160
Gly Glu Tyr Phe Ser Val Ala Pro Gln Asn Val His Ala Tyr Ile Ile
165 170 175
Gly Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser Gln Ala Asp Ile
180 185 190
Gly Gly Val Pro Ile Arg Lys Leu Val Glu Ser Lys Gly Glu Glu Ala
195 200 205
Gln Lys Glu Leu Glu Arg Ile Phe Val Asn Val Arg Asp Ala Ala Tyr
210 215 220
Gln Ile Ile Glu Lys Lys Gly Ala Thr Tyr Tyr Gly Ile Ala Met Gly
225 230 235 240
Leu Ala Arg Val Thr Arg Ala Ile Leu His Asn Glu Asn Ala Ile Leu
245 250 255
Thr Val Ser Ala Tyr Leu Asp Gly Pro Tyr Gly Glu Arg Asp Val Tyr
260 265 270
Ile Gly Val Pro Ala Val Ile Asn Arg Asn Gly Ile Arg Glu Val Ile
275 280 285
Glu Ile Glu Leu Asn Asp Asp Glu Lys Asn Arg Phe His His Ser Ala
290 295 300
Ala Thr Leu Lys Ser Val Leu Ala Arg Phe Phe Thr Arg
305 310 315
<210> 27
<211> 1002
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Actinomyces visosus)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(1002)
<400> 27
atg tcc gac cat atc acg aca acc gcc gag ggc tcc tac ccg acc aat 48
Met Ser Asp His Ile Thr Thr Thr Ala Glu Gly Ser Tyr Pro Thr Asn
1 5 10 15
cgg tcc gga cgg ccc agc aaa gtg gcc gtg atc ggc gcg ggc gcg gtc 96
Arg Ser Gly Arg Pro Ser Lys Val Ala Val Ile Gly Ala Gly Ala Val
20 25 30
ggg agc acc ctg gcc tac gcc tgt gtc acc aaa ggc gtc gct cgc gag 144
Gly Ser Thr Leu Ala Tyr Ala Cys Val Thr Lys Gly Val Ala Arg Glu
35 40 45
gtc gtc ttg cag gac ata gtc aag gaa aaa gta gaa gcc gaa gcg ctg 192
Val Val Leu Gln Asp Ile Val Lys Glu Lys Val Glu Ala Glu Ala Leu
50 55 60
gac atc gcc cag ggt atc cag ttc acc tcc gcg gga agt gtg tct ggc 240
Asp Ile Ala Gln Gly Ile Gln Phe Thr Ser Ala Gly Ser Val Ser Gly
65 70 75 80
tcg gat gac ccg gag atc tgc cgc gat gcc gac gtg att gcc atc act 288
Ser Asp Asp Pro Glu Ile Cys Arg Asp Ala Asp Val Ile Ala Ile Thr
85 90 95
gca ggc gca aag cag aag ccc ggc cag tcg cgc ttg gag ctg gcg gga 336
Ala Gly Ala Lys Gln Lys Pro Gly Gln Ser Arg Leu Glu Leu Ala Gly
100 105 110
gcg acc gtg ggg atc atg gaa aag atc ctg ccc aag ctc gtg gaa gtt 384
Ala Thr Val Gly Ile Met Glu Lys Ile Leu Pro Lys Leu Val Glu Val
115 120 125
gcc ccc aac gcc atc ttc gtc ctg gtc gcc aac ccg gtc gat gtg gtg 432
Ala Pro Asn Ala Ile Phe Val Leu Val Ala Asn Pro Val Asp Val Val
130 135 140
acg tac tgc gcc aag aaa atc acc ggc ctg ccg gag aac caa gtg ttt 480
Thr Tyr Cys Ala Lys Lys Ile Thr Gly Leu Pro Glu Asn Gln Val Phe
145 150 155 160
ggc tcg ggc act gtc ctc gac acg gcc cgt atg cgc tat ctg atc agc 528
Gly Ser Gly Thr Val Leu Asp Thr Ala Arg Met Arg Tyr Leu Ile Ser
165 170 175
ctc gaa acg ggg acc gct gtc cag aac atc cac ggc tat atc gcg ggt 576
Leu Glu Thr Gly Thr Ala Val Gln Asn Ile His Gly Tyr Ile Ala Gly
180 185 190
gag cat ggc gat tcg gaa gtc cca ctg tgg tcc tcg acc gaa atc ggc 624
Glu His Gly Asp Ser Glu Val Pro Leu Trp Ser Ser Thr Glu Ile Gly
195 200 205
ggt gtg ccg att acg cag tgg ggc acc acg ttg gac ggt ggc gtg ttc 672
Gly Val Pro Ile Thr Gln Trp Gly Thr Thr Leu Asp Gly Gly Val Phe
210 215 220
gac gag agc aag cgc gag cgg atc gcg cat gac gtc gtg cgc agc gcc 720
Asp Glu Ser Lys Arg Glu Arg Ile Ala His Asp Val Val Arg Ser Ala
225 230 235 240
tat cgg atc atc gag ggg aag ggc gcg acc aac tac gcg gtt ggc ctg 768
Tyr Arg Ile Ile Glu Gly Lys Gly Ala Thr Asn Tyr Ala Val Gly Leu
245 250 255
gcc gtg cag cgt atc atc ggc gca gtg ctg aat gac gag caa agg gtc 816
Ala Val Gln Arg Ile Ile Gly Ala Val Leu Asn Asp Glu Gln Arg Val
260 265 270
ctc acg atc agc ccg ctg ctc gat aac tgg cac ggc att tca gat gtg 864
Leu Thr Ile Ser Pro Leu Leu Asp Asn Trp His Gly Ile Ser Asp Val
275 280 285
tgc atg gcc gtc cct acc att gtt ggc cgc gag ggt gcg gga cgc cgg 912
Cys Met Ala Val Pro Thr Ile Val Gly Arg Glu Gly Ala Gly Arg Arg
290 295 300
ctg gag ctc ccg ctg acc ccc gaa gaa aag gaa cga ctg acc gct tcc 960
Leu Glu Leu Pro Leu Thr Pro Glu Glu Lys Glu Arg Leu Thr Ala Ser
305 310 315 320
gcc gac cac ctt cgg gaa gta gcc aga ggg ctg ggg tac taa 1002
Ala Asp His Leu Arg Glu Val Ala Arg Gly Leu Gly Tyr
325 330
<210> 28
<211> 333
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 28
Met Ser Asp His Ile Thr Thr Thr Ala Glu Gly Ser Tyr Pro Thr Asn
1 5 10 15
Arg Ser Gly Arg Pro Ser Lys Val Ala Val Ile Gly Ala Gly Ala Val
20 25 30
Gly Ser Thr Leu Ala Tyr Ala Cys Val Thr Lys Gly Val Ala Arg Glu
35 40 45
Val Val Leu Gln Asp Ile Val Lys Glu Lys Val Glu Ala Glu Ala Leu
50 55 60
Asp Ile Ala Gln Gly Ile Gln Phe Thr Ser Ala Gly Ser Val Ser Gly
65 70 75 80
Ser Asp Asp Pro Glu Ile Cys Arg Asp Ala Asp Val Ile Ala Ile Thr
85 90 95
Ala Gly Ala Lys Gln Lys Pro Gly Gln Ser Arg Leu Glu Leu Ala Gly
100 105 110
Ala Thr Val Gly Ile Met Glu Lys Ile Leu Pro Lys Leu Val Glu Val
115 120 125
Ala Pro Asn Ala Ile Phe Val Leu Val Ala Asn Pro Val Asp Val Val
130 135 140
Thr Tyr Cys Ala Lys Lys Ile Thr Gly Leu Pro Glu Asn Gln Val Phe
145 150 155 160
Gly Ser Gly Thr Val Leu Asp Thr Ala Arg Met Arg Tyr Leu Ile Ser
165 170 175
Leu Glu Thr Gly Thr Ala Val Gln Asn Ile His Gly Tyr Ile Ala Gly
180 185 190
Glu His Gly Asp Ser Glu Val Pro Leu Trp Ser Ser Thr Glu Ile Gly
195 200 205
Gly Val Pro Ile Thr Gln Trp Gly Thr Thr Leu Asp Gly Gly Val Phe
210 215 220
Asp Glu Ser Lys Arg Glu Arg Ile Ala His Asp Val Val Arg Ser Ala
225 230 235 240
Tyr Arg Ile Ile Glu Gly Lys Gly Ala Thr Asn Tyr Ala Val Gly Leu
245 250 255
Ala Val Gln Arg Ile Ile Gly Ala Val Leu Asn Asp Glu Gln Arg Val
260 265 270
Leu Thr Ile Ser Pro Leu Leu Asp Asn Trp His Gly Ile Ser Asp Val
275 280 285
Cys Met Ala Val Pro Thr Ile Val Gly Arg Glu Gly Ala Gly Arg Arg
290 295 300
Leu Glu Leu Pro Leu Thr Pro Glu Glu Lys Glu Arg Leu Thr Ala Ser
305 310 315 320
Ala Asp His Leu Arg Glu Val Ala Arg Gly Leu Gly Tyr
325 330
<210> 29
<211> 945
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Bacillus anthracis)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(945)
<400> 29
atg aaa aag ggc atc aac cga gtg gtc ctg gtc ggt acc gga gcc gtg 48
Met Lys Lys Gly Ile Asn Arg Val Val Leu Val Gly Thr Gly Ala Val
1 5 10 15
ggg tgc tcc tat gct tac tca atg atc aac cag ggc gtg gcc gag gaa 96
Gly Cys Ser Tyr Ala Tyr Ser Met Ile Asn Gln Gly Val Ala Glu Glu
20 25 30
ttc gtg ctg gtc gac gtc aac gaa gcc aag gcc gag ggt gag gca atg 144
Phe Val Leu Val Asp Val Asn Glu Ala Lys Ala Glu Gly Glu Ala Met
35 40 45
gac ctg tcc cac gcg gtc ccg ttc tcg ccc agc ccc acc aag gtg tgg 192
Asp Leu Ser His Ala Val Pro Phe Ser Pro Ser Pro Thr Lys Val Trp
50 55 60
agc ggc agc tac gcg gac tgc aaa gac gcc gat ctg gtg gtc atc acc 240
Ser Gly Ser Tyr Ala Asp Cys Lys Asp Ala Asp Leu Val Val Ile Thr
65 70 75 80
gcc gga ctc cct cag aag ccg ggt gaa acc cgc ttg gac ctg gtc gag 288
Ala Gly Leu Pro Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val Glu
85 90 95
aag aac acg aag atc ttc aaa cag atc gtg agg gga atc atg gat tcc 336
Lys Asn Thr Lys Ile Phe Lys Gln Ile Val Arg Gly Ile Met Asp Ser
100 105 110
ggc ttt gac ggc atc ttc ctc atc gcg acg aac ccc gtg gat atc ttg 384
Gly Phe Asp Gly Ile Phe Leu Ile Ala Thr Asn Pro Val Asp Ile Leu
115 120 125
acc tat gtc acc tgg aag gaa tcc ggc ctc ccg aaa gag cgc gtc att 432
Thr Tyr Val Thr Trp Lys Glu Ser Gly Leu Pro Lys Glu Arg Val Ile
130 135 140
ggc agt ggg acc acg ctg gac agc gct cgc ttc cgg tat atg ctg ggc 480
Gly Ser Gly Thr Thr Leu Asp Ser Ala Arg Phe Arg Tyr Met Leu Gly
145 150 155 160
gac tac ctg gac gtg gac ccg cgg aac gtc cat gcc tac atc gta ggt 528
Asp Tyr Leu Asp Val Asp Pro Arg Asn Val His Ala Tyr Ile Val Gly
165 170 175
gaa cat ggc gat acc gag ctg ccc gtt tgg tcg cac gcg acg atc ggc 576
Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser His Ala Thr Ile Gly
180 185 190
gtg cag aag ctg gaa aca atc ctc gcc aac aat gaa cag tac aag caa 624
Val Gln Lys Leu Glu Thr Ile Leu Ala Asn Asn Glu Gln Tyr Lys Gln
195 200 205
gag gat ctc gat aag att ttc gaa aac gtc cgg gac gcg gcc tac cat 672
Glu Asp Leu Asp Lys Ile Phe Glu Asn Val Arg Asp Ala Ala Tyr His
210 215 220
atc atc gag cgc aaa ggg gcc acc tat tac ggc atc ggg atg tcg ctc 720
Ile Ile Glu Arg Lys Gly Ala Thr Tyr Tyr Gly Ile Gly Met Ser Leu
225 230 235 240
ctg cgg gtg acc aag gca ata ctg aat aat gag aat agc gtg ctg act 768
Leu Arg Val Thr Lys Ala Ile Leu Asn Asn Glu Asn Ser Val Leu Thr
245 250 255
gtc tct gcg tac ctt gag ggc cag tat ggc gag aaa gac gcg tac gtg 816
Val Ser Ala Tyr Leu Glu Gly Gln Tyr Gly Glu Lys Asp Ala Tyr Val
260 265 270
ggc gta ccg gcc gtt atc aac cgc gag ggc gtc cgt gaa atc gtc gaa 864
Gly Val Pro Ala Val Ile Asn Arg Glu Gly Val Arg Glu Ile Val Glu
275 280 285
ctg gag ttg aac gaa gag gaa aag gcc aag ttc gcc cac tcg gtg aag 912
Leu Glu Leu Asn Glu Glu Glu Lys Ala Lys Phe Ala His Ser Val Lys
290 295 300
gtc ctc aag gaa acg atg gcg cca gtg ctg taa 945
Val Leu Lys Glu Thr Met Ala Pro Val Leu
305 310
<210> 30
<211> 314
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 30
Met Lys Lys Gly Ile Asn Arg Val Val Leu Val Gly Thr Gly Ala Val
1 5 10 15
Gly Cys Ser Tyr Ala Tyr Ser Met Ile Asn Gln Gly Val Ala Glu Glu
20 25 30
Phe Val Leu Val Asp Val Asn Glu Ala Lys Ala Glu Gly Glu Ala Met
35 40 45
Asp Leu Ser His Ala Val Pro Phe Ser Pro Ser Pro Thr Lys Val Trp
50 55 60
Ser Gly Ser Tyr Ala Asp Cys Lys Asp Ala Asp Leu Val Val Ile Thr
65 70 75 80
Ala Gly Leu Pro Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val Glu
85 90 95
Lys Asn Thr Lys Ile Phe Lys Gln Ile Val Arg Gly Ile Met Asp Ser
100 105 110
Gly Phe Asp Gly Ile Phe Leu Ile Ala Thr Asn Pro Val Asp Ile Leu
115 120 125
Thr Tyr Val Thr Trp Lys Glu Ser Gly Leu Pro Lys Glu Arg Val Ile
130 135 140
Gly Ser Gly Thr Thr Leu Asp Ser Ala Arg Phe Arg Tyr Met Leu Gly
145 150 155 160
Asp Tyr Leu Asp Val Asp Pro Arg Asn Val His Ala Tyr Ile Val Gly
165 170 175
Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser His Ala Thr Ile Gly
180 185 190
Val Gln Lys Leu Glu Thr Ile Leu Ala Asn Asn Glu Gln Tyr Lys Gln
195 200 205
Glu Asp Leu Asp Lys Ile Phe Glu Asn Val Arg Asp Ala Ala Tyr His
210 215 220
Ile Ile Glu Arg Lys Gly Ala Thr Tyr Tyr Gly Ile Gly Met Ser Leu
225 230 235 240
Leu Arg Val Thr Lys Ala Ile Leu Asn Asn Glu Asn Ser Val Leu Thr
245 250 255
Val Ser Ala Tyr Leu Glu Gly Gln Tyr Gly Glu Lys Asp Ala Tyr Val
260 265 270
Gly Val Pro Ala Val Ile Asn Arg Glu Gly Val Arg Glu Ile Val Glu
275 280 285
Leu Glu Leu Asn Glu Glu Glu Lys Ala Lys Phe Ala His Ser Val Lys
290 295 300
Val Leu Lys Glu Thr Met Ala Pro Val Leu
305 310
<210> 31
<211> 957
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Ruminococcus torques)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(957)
<400> 31
atg aag aaa aaa gtg atc aac tcg aag aaa gcc gtg atg atc ggg tgc 48
Met Lys Lys Lys Val Ile Asn Ser Lys Lys Ala Val Met Ile Gly Cys
1 5 10 15
ggc ttt gtg ggt agc gcg tcg gtg ttc gca ctg atg cag agt ggc ctg 96
Gly Phe Val Gly Ser Ala Ser Val Phe Ala Leu Met Gln Ser Gly Leu
20 25 30
ttc acc gag atc gtc ctc atc gac gct gac aag aat aag gcc gaa ggc 144
Phe Thr Glu Ile Val Leu Ile Asp Ala Asp Lys Asn Lys Ala Glu Gly
35 40 45
gag gcc atg gat atc tcg cac ggt att ccg ttc gcg tcc cct atg aag 192
Glu Ala Met Asp Ile Ser His Gly Ile Pro Phe Ala Ser Pro Met Lys
50 55 60
atc tat gcg ggc gac tac gat gat gtc gcc gat gcg gca atc gtt gtg 240
Ile Tyr Ala Gly Asp Tyr Asp Asp Val Ala Asp Ala Ala Ile Val Val
65 70 75 80
atc tcc gcc gga gcc ggc cag aaa ccc ggc gag act cgg ctg gac ctg 288
Ile Ser Ala Gly Ala Gly Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu
85 90 95
gtc aac aaa aac gtc gcg atc ttc aag tcg atc att ccg gag att gcg 336
Val Asn Lys Asn Val Ala Ile Phe Lys Ser Ile Ile Pro Glu Ile Ala
100 105 110
aag cgc aac ttc gcc ggt atc atg ctg gtc gtc gcc aac ccg gtc gat 384
Lys Arg Asn Phe Ala Gly Ile Met Leu Val Val Ala Asn Pro Val Asp
115 120 125
atc ctg acc caa gtg gcc atc aag ttg tcc ggg ctg ccc gag aac cgc 432
Ile Leu Thr Gln Val Ala Ile Lys Leu Ser Gly Leu Pro Glu Asn Arg
130 135 140
gtc atc ggc tcg ggc acg gtg ctc gac agt gcg cgg ctg agg tac aag 480
Val Ile Gly Ser Gly Thr Val Leu Asp Ser Ala Arg Leu Arg Tyr Lys
145 150 155 160
ttg ggg gaa cat ctc tcc gtc gac tcc cgg agc gtg cat gcc ttc atc 528
Leu Gly Glu His Leu Ser Val Asp Ser Arg Ser Val His Ala Phe Ile
165 170 175
gtc ggc gaa cac ggg gac tcc gaa gtc gtg gct tgg agc agc gcg aat 576
Val Gly Glu His Gly Asp Ser Glu Val Val Ala Trp Ser Ser Ala Asn
180 185 190
gtg tcg gga gtg ccg ctg agc gaa atg tgc gag atg cgt ggc cac tac 624
Val Ser Gly Val Pro Leu Ser Glu Met Cys Glu Met Arg Gly His Tyr
195 200 205
aag cat aag gaa aac acg gcc gaa atc gcc acc gcg gta aag aat tct 672
Lys His Lys Glu Asn Thr Ala Glu Ile Ala Thr Ala Val Lys Asn Ser
210 215 220
gcg tac gag ata atc aac aag aag cac gcc acc tac tat ggc atc gca 720
Ala Tyr Glu Ile Ile Asn Lys Lys His Ala Thr Tyr Tyr Gly Ile Ala
225 230 235 240
atg agc gtt aaa cgc atc tgt gag gtc atc atg cgc gat gag aag tcg 768
Met Ser Val Lys Arg Ile Cys Glu Val Ile Met Arg Asp Glu Lys Ser
245 250 255
atc ctt ccc gta tcc cac atg att cat ggc gtc tat gac atc gac ggc 816
Ile Leu Pro Val Ser His Met Ile His Gly Val Tyr Asp Ile Asp Gly
260 265 270
gtg agc ctc tca atg cca gcc atc gtg ggt gcc gac ggc atc gag agc 864
Val Ser Leu Ser Met Pro Ala Ile Val Gly Ala Asp Gly Ile Glu Ser
275 280 285
gac atc ccg ata aac ctc agc gga gaa gaa gcg ctg aag ctg aaa gag 912
Asp Ile Pro Ile Asn Leu Ser Gly Glu Glu Ala Leu Lys Leu Lys Glu
290 295 300
tcc gcc gac tcg ctg aag aaa atc atc gaa acc atc gaa ctg taa 957
Ser Ala Asp Ser Leu Lys Lys Ile Ile Glu Thr Ile Glu Leu
305 310 315
<210> 32
<211> 318
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 32
Met Lys Lys Lys Val Ile Asn Ser Lys Lys Ala Val Met Ile Gly Cys
1 5 10 15
Gly Phe Val Gly Ser Ala Ser Val Phe Ala Leu Met Gln Ser Gly Leu
20 25 30
Phe Thr Glu Ile Val Leu Ile Asp Ala Asp Lys Asn Lys Ala Glu Gly
35 40 45
Glu Ala Met Asp Ile Ser His Gly Ile Pro Phe Ala Ser Pro Met Lys
50 55 60
Ile Tyr Ala Gly Asp Tyr Asp Asp Val Ala Asp Ala Ala Ile Val Val
65 70 75 80
Ile Ser Ala Gly Ala Gly Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu
85 90 95
Val Asn Lys Asn Val Ala Ile Phe Lys Ser Ile Ile Pro Glu Ile Ala
100 105 110
Lys Arg Asn Phe Ala Gly Ile Met Leu Val Val Ala Asn Pro Val Asp
115 120 125
Ile Leu Thr Gln Val Ala Ile Lys Leu Ser Gly Leu Pro Glu Asn Arg
130 135 140
Val Ile Gly Ser Gly Thr Val Leu Asp Ser Ala Arg Leu Arg Tyr Lys
145 150 155 160
Leu Gly Glu His Leu Ser Val Asp Ser Arg Ser Val His Ala Phe Ile
165 170 175
Val Gly Glu His Gly Asp Ser Glu Val Val Ala Trp Ser Ser Ala Asn
180 185 190
Val Ser Gly Val Pro Leu Ser Glu Met Cys Glu Met Arg Gly His Tyr
195 200 205
Lys His Lys Glu Asn Thr Ala Glu Ile Ala Thr Ala Val Lys Asn Ser
210 215 220
Ala Tyr Glu Ile Ile Asn Lys Lys His Ala Thr Tyr Tyr Gly Ile Ala
225 230 235 240
Met Ser Val Lys Arg Ile Cys Glu Val Ile Met Arg Asp Glu Lys Ser
245 250 255
Ile Leu Pro Val Ser His Met Ile His Gly Val Tyr Asp Ile Asp Gly
260 265 270
Val Ser Leu Ser Met Pro Ala Ile Val Gly Ala Asp Gly Ile Glu Ser
275 280 285
Asp Ile Pro Ile Asn Leu Ser Gly Glu Glu Ala Leu Lys Leu Lys Glu
290 295 300
Ser Ala Asp Ser Leu Lys Lys Ile Ile Glu Thr Ile Glu Leu
305 310 315
<210> 33
<211> 942
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Listeria marthii)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(942)
<400> 33
atg aag gat cat cag aag atc atc ctg gta ggc gat ggt gcc gtg gga 48
Met Lys Asp His Gln Lys Ile Ile Leu Val Gly Asp Gly Ala Val Gly
1 5 10 15
tcc tcc tac gcc ttc gcc tgc gtc aac ctt tcg att ggc cag gaa ttc 96
Ser Ser Tyr Ala Phe Ala Cys Val Asn Leu Ser Ile Gly Gln Glu Phe
20 25 30
ggc att atc gac atc gac aag gat agg aca atc ggt gac gcg atg gac 144
Gly Ile Ile Asp Ile Asp Lys Asp Arg Thr Ile Gly Asp Ala Met Asp
35 40 45
ctc tcc cat gcc gtg ccc ttc tct acc ccg aaa aag atc tac tcc gcg 192
Leu Ser His Ala Val Pro Phe Ser Thr Pro Lys Lys Ile Tyr Ser Ala
50 55 60
aac tat tcg gac tgc cac gac gcg gat ctg gtg gtc gtc acg gct ggg 240
Asn Tyr Ser Asp Cys His Asp Ala Asp Leu Val Val Val Thr Ala Gly
65 70 75 80
acc gcc cag aag cct ggc gaa acg cgc ctg gac ttg gtt aac cgc aat 288
Thr Ala Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val Asn Arg Asn
85 90 95
atc aag atc atg aag ggc ata gtg gac gaa gtc atg gcg agc ggt ttt 336
Ile Lys Ile Met Lys Gly Ile Val Asp Glu Val Met Ala Ser Gly Phe
100 105 110
gac ggc atc ttc ctg atc gcg agc aac ccc gtg gac atc ctg acc tac 384
Asp Gly Ile Phe Leu Ile Ala Ser Asn Pro Val Asp Ile Leu Thr Tyr
115 120 125
gcg acc tgg aag ttc agc ggg ctc ccc aaa gag cgg gtc atc ggc tcg 432
Ala Thr Trp Lys Phe Ser Gly Leu Pro Lys Glu Arg Val Ile Gly Ser
130 135 140
gga acc tca ctc gat acc gca cgc ttc cgg atg agt atc gcc gac tac 480
Gly Thr Ser Leu Asp Thr Ala Arg Phe Arg Met Ser Ile Ala Asp Tyr
145 150 155 160
ctg aag gtc gat gcg cgg aat gtt cac ggc tac atc ctg ggg gag cac 528
Leu Lys Val Asp Ala Arg Asn Val His Gly Tyr Ile Leu Gly Glu His
165 170 175
ggg gat acg gaa ttc ccg gcc tgg agc cac acc act gtc ggc ggc ttg 576
Gly Asp Thr Glu Phe Pro Ala Trp Ser His Thr Thr Val Gly Gly Leu
180 185 190
ccg atc acc gag tgg atc tcg gaa gat gaa cag ggc gcc atg gac acg 624
Pro Ile Thr Glu Trp Ile Ser Glu Asp Glu Gln Gly Ala Met Asp Thr
195 200 205
atc ttc gtg tcc gtg cgt gat gcc gcc tac gag atc atc aac aaa aag 672
Ile Phe Val Ser Val Arg Asp Ala Ala Tyr Glu Ile Ile Asn Lys Lys
210 215 220
gga gcc acc ttc tat ggc gtc gcg gca gcc ctc gcc cgc atc acg aag 720
Gly Ala Thr Phe Tyr Gly Val Ala Ala Ala Leu Ala Arg Ile Thr Lys
225 230 235 240
gcc att ctg aat aac gag aac gcg atc ctg ccg ctg tcg gtc tac ctg 768
Ala Ile Leu Asn Asn Glu Asn Ala Ile Leu Pro Leu Ser Val Tyr Leu
245 250 255
gac ggc cat tat ggc atg aac gac atc tat atc ggc gct ccg gcg gtc 816
Asp Gly His Tyr Gly Met Asn Asp Ile Tyr Ile Gly Ala Pro Ala Val
260 265 270
gtg aat cgg cag ggt gtg cga cac atc gtg gag atg aac ctc acc gac 864
Val Asn Arg Gln Gly Val Arg His Ile Val Glu Met Asn Leu Thr Asp
275 280 285
aaa gaa aag gaa cag atg aag aac agc gcg gac acc ctg aaa aag gtc 912
Lys Glu Lys Glu Gln Met Lys Asn Ser Ala Asp Thr Leu Lys Lys Val
290 295 300
ctg gac gac gcc atg aaa caa gtg gac taa 942
Leu Asp Asp Ala Met Lys Gln Val Asp
305 310
<210> 34
<211> 313
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 34
Met Lys Asp His Gln Lys Ile Ile Leu Val Gly Asp Gly Ala Val Gly
1 5 10 15
Ser Ser Tyr Ala Phe Ala Cys Val Asn Leu Ser Ile Gly Gln Glu Phe
20 25 30
Gly Ile Ile Asp Ile Asp Lys Asp Arg Thr Ile Gly Asp Ala Met Asp
35 40 45
Leu Ser His Ala Val Pro Phe Ser Thr Pro Lys Lys Ile Tyr Ser Ala
50 55 60
Asn Tyr Ser Asp Cys His Asp Ala Asp Leu Val Val Val Thr Ala Gly
65 70 75 80
Thr Ala Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val Asn Arg Asn
85 90 95
Ile Lys Ile Met Lys Gly Ile Val Asp Glu Val Met Ala Ser Gly Phe
100 105 110
Asp Gly Ile Phe Leu Ile Ala Ser Asn Pro Val Asp Ile Leu Thr Tyr
115 120 125
Ala Thr Trp Lys Phe Ser Gly Leu Pro Lys Glu Arg Val Ile Gly Ser
130 135 140
Gly Thr Ser Leu Asp Thr Ala Arg Phe Arg Met Ser Ile Ala Asp Tyr
145 150 155 160
Leu Lys Val Asp Ala Arg Asn Val His Gly Tyr Ile Leu Gly Glu His
165 170 175
Gly Asp Thr Glu Phe Pro Ala Trp Ser His Thr Thr Val Gly Gly Leu
180 185 190
Pro Ile Thr Glu Trp Ile Ser Glu Asp Glu Gln Gly Ala Met Asp Thr
195 200 205
Ile Phe Val Ser Val Arg Asp Ala Ala Tyr Glu Ile Ile Asn Lys Lys
210 215 220
Gly Ala Thr Phe Tyr Gly Val Ala Ala Ala Leu Ala Arg Ile Thr Lys
225 230 235 240
Ala Ile Leu Asn Asn Glu Asn Ala Ile Leu Pro Leu Ser Val Tyr Leu
245 250 255
Asp Gly His Tyr Gly Met Asn Asp Ile Tyr Ile Gly Ala Pro Ala Val
260 265 270
Val Asn Arg Gln Gly Val Arg His Ile Val Glu Met Asn Leu Thr Asp
275 280 285
Lys Glu Lys Glu Gln Met Lys Asn Ser Ala Asp Thr Leu Lys Lys Val
290 295 300
Leu Asp Asp Ala Met Lys Gln Val Asp
305 310
<210> 35
<211> 963
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Bacillus subtilis)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(963)
<400> 35
atg aac aaa cac gtg aat aaa gtg gcc ctg atc ggc gca ggc ttc gtc 48
Met Asn Lys His Val Asn Lys Val Ala Leu Ile Gly Ala Gly Phe Val
1 5 10 15
ggg agc tcg tac gcg ttc gcg ctg atc aac cag ggc atc acc gat gag 96
Gly Ser Ser Tyr Ala Phe Ala Leu Ile Asn Gln Gly Ile Thr Asp Glu
20 25 30
ctg gta gtc atc gat gtt aac aaa gaa aag gcc atg ggg gat gtg atg 144
Leu Val Val Ile Asp Val Asn Lys Glu Lys Ala Met Gly Asp Val Met
35 40 45
gac ttg aat cac ggc aag gcc ttt gcc ccg cag ccc gtc aag acg agc 192
Asp Leu Asn His Gly Lys Ala Phe Ala Pro Gln Pro Val Lys Thr Ser
50 55 60
tat ggg acg tac gaa gat tgt aaa gac gcc gac atc gtg tgc atc tgc 240
Tyr Gly Thr Tyr Glu Asp Cys Lys Asp Ala Asp Ile Val Cys Ile Cys
65 70 75 80
gct ggc gcc aat cag aag ccg gga gag act cgc ctg gag ctg gtc gag 288
Ala Gly Ala Asn Gln Lys Pro Gly Glu Thr Arg Leu Glu Leu Val Glu
85 90 95
aag aac ctg aaa atc ttc aag ggc att gtg tcc gaa gtg atg gcc agc 336
Lys Asn Leu Lys Ile Phe Lys Gly Ile Val Ser Glu Val Met Ala Ser
100 105 110
ggc ttc gac ggc atc ttc ctt atc gcc aca aac ccg gtc gac atc ctg 384
Gly Phe Asp Gly Ile Phe Leu Ile Ala Thr Asn Pro Val Asp Ile Leu
115 120 125
acc tat gcc acc tgg aag ttc tcg ggc ctc cct aag gaa cgc gtc atc 432
Thr Tyr Ala Thr Trp Lys Phe Ser Gly Leu Pro Lys Glu Arg Val Ile
130 135 140
gga tct ggc acc acg ctc gac tcg gcg cgt ttc cgc tac atg ctc tcc 480
Gly Ser Gly Thr Thr Leu Asp Ser Ala Arg Phe Arg Tyr Met Leu Ser
145 150 155 160
gaa tac ttc ggc gct gcg ccc caa aac gtc cat gcc cac atc ata ggc 528
Glu Tyr Phe Gly Ala Ala Pro Gln Asn Val His Ala His Ile Ile Gly
165 170 175
gag cac ggc gac acc gag ctg ccg gtc tgg tcc cac gcg aac gtg ggt 576
Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser His Ala Asn Val Gly
180 185 190
ggc gtc ccc gtg tcg gag ttg gtg gag aag aat gac gcg tat aag caa 624
Gly Val Pro Val Ser Glu Leu Val Glu Lys Asn Asp Ala Tyr Lys Gln
195 200 205
gaa gaa ctg gac cag atc gtc gat gac gtg aag aac gcg gcc tac cat 672
Glu Glu Leu Asp Gln Ile Val Asp Asp Val Lys Asn Ala Ala Tyr His
210 215 220
atc att gag aag aaa ggc gcg acc tac tac ggt gtc gca atg tcc ctg 720
Ile Ile Glu Lys Lys Gly Ala Thr Tyr Tyr Gly Val Ala Met Ser Leu
225 230 235 240
gcg cgg atc acc aag gcc atc ctg cat aac gag aat tca atc ctc acg 768
Ala Arg Ile Thr Lys Ala Ile Leu His Asn Glu Asn Ser Ile Leu Thr
245 250 255
gtg agt acc tat ttg gac ggc cag tat ggg gcc gac gat gtc tac atc 816
Val Ser Thr Tyr Leu Asp Gly Gln Tyr Gly Ala Asp Asp Val Tyr Ile
260 265 270
gga gtt cca gcg gta gtc aac cgg ggt ggc atc gcc ggg atc acc gaa 864
Gly Val Pro Ala Val Val Asn Arg Gly Gly Ile Ala Gly Ile Thr Glu
275 280 285
ctc aac ctg aac gaa aag gaa aag gaa cag ttc ctg cac agc gcc ggt 912
Leu Asn Leu Asn Glu Lys Glu Lys Glu Gln Phe Leu His Ser Ala Gly
290 295 300
gtg ctg aag aac att ctg aag ccg cat ttt gcc gag cag aaa gtg aac 960
Val Leu Lys Asn Ile Leu Lys Pro His Phe Ala Glu Gln Lys Val Asn
305 310 315 320
taa 963
<210> 36
<211> 320
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 36
Met Asn Lys His Val Asn Lys Val Ala Leu Ile Gly Ala Gly Phe Val
1 5 10 15
Gly Ser Ser Tyr Ala Phe Ala Leu Ile Asn Gln Gly Ile Thr Asp Glu
20 25 30
Leu Val Val Ile Asp Val Asn Lys Glu Lys Ala Met Gly Asp Val Met
35 40 45
Asp Leu Asn His Gly Lys Ala Phe Ala Pro Gln Pro Val Lys Thr Ser
50 55 60
Tyr Gly Thr Tyr Glu Asp Cys Lys Asp Ala Asp Ile Val Cys Ile Cys
65 70 75 80
Ala Gly Ala Asn Gln Lys Pro Gly Glu Thr Arg Leu Glu Leu Val Glu
85 90 95
Lys Asn Leu Lys Ile Phe Lys Gly Ile Val Ser Glu Val Met Ala Ser
100 105 110
Gly Phe Asp Gly Ile Phe Leu Ile Ala Thr Asn Pro Val Asp Ile Leu
115 120 125
Thr Tyr Ala Thr Trp Lys Phe Ser Gly Leu Pro Lys Glu Arg Val Ile
130 135 140
Gly Ser Gly Thr Thr Leu Asp Ser Ala Arg Phe Arg Tyr Met Leu Ser
145 150 155 160
Glu Tyr Phe Gly Ala Ala Pro Gln Asn Val His Ala His Ile Ile Gly
165 170 175
Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser His Ala Asn Val Gly
180 185 190
Gly Val Pro Val Ser Glu Leu Val Glu Lys Asn Asp Ala Tyr Lys Gln
195 200 205
Glu Glu Leu Asp Gln Ile Val Asp Asp Val Lys Asn Ala Ala Tyr His
210 215 220
Ile Ile Glu Lys Lys Gly Ala Thr Tyr Tyr Gly Val Ala Met Ser Leu
225 230 235 240
Ala Arg Ile Thr Lys Ala Ile Leu His Asn Glu Asn Ser Ile Leu Thr
245 250 255
Val Ser Thr Tyr Leu Asp Gly Gln Tyr Gly Ala Asp Asp Val Tyr Ile
260 265 270
Gly Val Pro Ala Val Val Asn Arg Gly Gly Ile Ala Gly Ile Thr Glu
275 280 285
Leu Asn Leu Asn Glu Lys Glu Lys Glu Gln Phe Leu His Ser Ala Gly
290 295 300
Val Leu Lys Asn Ile Leu Lys Pro His Phe Ala Glu Gln Lys Val Asn
305 310 315 320
<210> 37
<211> 945
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Enterococcus faecium)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(945)
<400> 37
atg aag aaa acg agc cgc aaa gtc gtg atc gtc ggg acc ggc ttc gtc 48
Met Lys Lys Thr Ser Arg Lys Val Val Ile Val Gly Thr Gly Phe Val
1 5 10 15
ggc acc tcc atc gct tac gcc atg atc aat cag ggt att tcg aac gaa 96
Gly Thr Ser Ile Ala Tyr Ala Met Ile Asn Gln Gly Ile Ser Asn Glu
20 25 30
ctg gtg ctg atc gac gtg aat cag gaa aag gcc gaa ggt gag gca ctg 144
Leu Val Leu Ile Asp Val Asn Gln Glu Lys Ala Glu Gly Glu Ala Leu
35 40 45
gat ctg ctg gat ggc atg gcc tgg ggc gac gag aac gtg gcg gtc tgg 192
Asp Leu Leu Asp Gly Met Ala Trp Gly Asp Glu Asn Val Ala Val Trp
50 55 60
agc ggt ggc tat gaa gag tgc aaa gac gcc gac att gtg gtc gtc acc 240
Ser Gly Gly Tyr Glu Glu Cys Lys Asp Ala Asp Ile Val Val Val Thr
65 70 75 80
gcc ggt atc aac caa aag ccg gga cag tct agg ttg gat ctg gtc aag 288
Ala Gly Ile Asn Gln Lys Pro Gly Gln Ser Arg Leu Asp Leu Val Lys
85 90 95
acg aat gcc tcc atc atg cgg cag atc gtc aaa gaa atc atg ggg tcc 336
Thr Asn Ala Ser Ile Met Arg Gln Ile Val Lys Glu Ile Met Gly Ser
100 105 110
gga ttc gac ggc atc atc gtg gtg gcg tcg aac cct gta gac atc ctc 384
Gly Phe Asp Gly Ile Ile Val Val Ala Ser Asn Pro Val Asp Ile Leu
115 120 125
acc tac atc gcg tgg aac gag tcc ggg ctg ccg acg agc cgg gtg atc 432
Thr Tyr Ile Ala Trp Asn Glu Ser Gly Leu Pro Thr Ser Arg Val Ile
130 135 140
ggg acg ggc acg acc ctg gac acc acc cgg ttc cgg aag gaa atc gcg 480
Gly Thr Gly Thr Thr Leu Asp Thr Thr Arg Phe Arg Lys Glu Ile Ala
145 150 155 160
ctc aag ctc aag gtc gac ccc cgc tcg gtc cat ggc tat atc ctg ggc 528
Leu Lys Leu Lys Val Asp Pro Arg Ser Val His Gly Tyr Ile Leu Gly
165 170 175
gag cat ggc gac agc gaa gtg gcg gcc tgg agc cac aca acg gtg ggc 576
Glu His Gly Asp Ser Glu Val Ala Ala Trp Ser His Thr Thr Val Gly
180 185 190
ggc aag ccc gtc ttt gag atc gtt gaa aag gac cac cgt atc gcg aaa 624
Gly Lys Pro Val Phe Glu Ile Val Glu Lys Asp His Arg Ile Ala Lys
195 200 205
gac gag ttg gat gtc ata gcc gat aag gtc cgc aac gcg gcc tat gaa 672
Asp Glu Leu Asp Val Ile Ala Asp Lys Val Arg Asn Ala Ala Tyr Glu
210 215 220
atc atc gac cgc aag aaa gcc acc tac tac ggg atc ggc atg tcg acc 720
Ile Ile Asp Arg Lys Lys Ala Thr Tyr Tyr Gly Ile Gly Met Ser Thr
225 230 235 240
gcg cgg atc gtg aag gcc ata ctg aat aac gaa cag gcc gtg ctg ccg 768
Ala Arg Ile Val Lys Ala Ile Leu Asn Asn Glu Gln Ala Val Leu Pro
245 250 255
gtt tca gct tac ctg acc gga gag tac aac gag aaa gac atc ttc acc 816
Val Ser Ala Tyr Leu Thr Gly Glu Tyr Asn Glu Lys Asp Ile Phe Thr
260 265 270
ggc gtt ccg tcg atc gtg gat gag aac ggc gtg cga gaa gtc gtg gag 864
Gly Val Pro Ser Ile Val Asp Glu Asn Gly Val Arg Glu Val Val Glu
275 280 285
ctc agt att aac gaa gaa gag aaa gcg atg ttc tcc aag agc acc tcc 912
Leu Ser Ile Asn Glu Glu Glu Lys Ala Met Phe Ser Lys Ser Thr Ser
290 295 300
gca ctt cgc gag gta ctg aac act gtc ctc taa 945
Ala Leu Arg Glu Val Leu Asn Thr Val Leu
305 310
<210> 38
<211> 314
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 38
Met Lys Lys Thr Ser Arg Lys Val Val Ile Val Gly Thr Gly Phe Val
1 5 10 15
Gly Thr Ser Ile Ala Tyr Ala Met Ile Asn Gln Gly Ile Ser Asn Glu
20 25 30
Leu Val Leu Ile Asp Val Asn Gln Glu Lys Ala Glu Gly Glu Ala Leu
35 40 45
Asp Leu Leu Asp Gly Met Ala Trp Gly Asp Glu Asn Val Ala Val Trp
50 55 60
Ser Gly Gly Tyr Glu Glu Cys Lys Asp Ala Asp Ile Val Val Val Thr
65 70 75 80
Ala Gly Ile Asn Gln Lys Pro Gly Gln Ser Arg Leu Asp Leu Val Lys
85 90 95
Thr Asn Ala Ser Ile Met Arg Gln Ile Val Lys Glu Ile Met Gly Ser
100 105 110
Gly Phe Asp Gly Ile Ile Val Val Ala Ser Asn Pro Val Asp Ile Leu
115 120 125
Thr Tyr Ile Ala Trp Asn Glu Ser Gly Leu Pro Thr Ser Arg Val Ile
130 135 140
Gly Thr Gly Thr Thr Leu Asp Thr Thr Arg Phe Arg Lys Glu Ile Ala
145 150 155 160
Leu Lys Leu Lys Val Asp Pro Arg Ser Val His Gly Tyr Ile Leu Gly
165 170 175
Glu His Gly Asp Ser Glu Val Ala Ala Trp Ser His Thr Thr Val Gly
180 185 190
Gly Lys Pro Val Phe Glu Ile Val Glu Lys Asp His Arg Ile Ala Lys
195 200 205
Asp Glu Leu Asp Val Ile Ala Asp Lys Val Arg Asn Ala Ala Tyr Glu
210 215 220
Ile Ile Asp Arg Lys Lys Ala Thr Tyr Tyr Gly Ile Gly Met Ser Thr
225 230 235 240
Ala Arg Ile Val Lys Ala Ile Leu Asn Asn Glu Gln Ala Val Leu Pro
245 250 255
Val Ser Ala Tyr Leu Thr Gly Glu Tyr Asn Glu Lys Asp Ile Phe Thr
260 265 270
Gly Val Pro Ser Ile Val Asp Glu Asn Gly Val Arg Glu Val Val Glu
275 280 285
Leu Ser Ile Asn Glu Glu Glu Lys Ala Met Phe Ser Lys Ser Thr Ser
290 295 300
Ala Leu Arg Glu Val Leu Asn Thr Val Leu
305 310
<210> 39
<211> 951
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Bacillus thuriengensis)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(951)
<400> 39
atg aaa cgg cat acc cgg aaa ata gcc atc atc ggc acg ggc ctg gtg 48
Met Lys Arg His Thr Arg Lys Ile Ala Ile Ile Gly Thr Gly Leu Val
1 5 10 15
ggg tcc agc tgc gca tac tcc atc gtc aac cag ggt atc tgc gag gaa 96
Gly Ser Ser Cys Ala Tyr Ser Ile Val Asn Gln Gly Ile Cys Glu Glu
20 25 30
ctc ctg ctg atc gac atc aac cac gag cgc gct gtc ggc gag gcg atg 144
Leu Leu Leu Ile Asp Ile Asn His Glu Arg Ala Val Gly Glu Ala Met
35 40 45
gat ctc tcc cat tgc atc aac ttc acg aac acg cgc acc aag gtg tac 192
Asp Leu Ser His Cys Ile Asn Phe Thr Asn Thr Arg Thr Lys Val Tyr
50 55 60
gcg ggg tcg tac gaa gat tgt aaa gac atg gac atc gtt atc att acc 240
Ala Gly Ser Tyr Glu Asp Cys Lys Asp Met Asp Ile Val Ile Ile Thr
65 70 75 80
gcg ggt ccg gca ccc aag cct ggc cag tcg cgg ctg gat acg ttg ggc 288
Ala Gly Pro Ala Pro Lys Pro Gly Gln Ser Arg Leu Asp Thr Leu Gly
85 90 95
gcc tcc gcc aag atc atg gag agc gtt gtg ggt gga gtc atg gaa tcg 336
Ala Ser Ala Lys Ile Met Glu Ser Val Val Gly Gly Val Met Glu Ser
100 105 110
ggg ttt gac ggc atc ttc ctt ctc gcc agt aat ccg gtg gac atc atc 384
Gly Phe Asp Gly Ile Phe Leu Leu Ala Ser Asn Pro Val Asp Ile Ile
115 120 125
acc tat caa gtc tgg aag ttg tcg ggc ctc ccc agg aat cga gtg atc 432
Thr Tyr Gln Val Trp Lys Leu Ser Gly Leu Pro Arg Asn Arg Val Ile
130 135 140
ggg acc ggc acc agc ctg gac agc tca cgg ctg cgt acc atc ctg tcg 480
Gly Thr Gly Thr Ser Leu Asp Ser Ser Arg Leu Arg Thr Ile Leu Ser
145 150 155 160
gaa atg ctg cac gtc gat ccg cgc agc atc cac ggc tac agc ctc ggc 528
Glu Met Leu His Val Asp Pro Arg Ser Ile His Gly Tyr Ser Leu Gly
165 170 175
gag cac ggc gac tcg cag atg gtc gcc tgg agt cac gtc acc gtc ggc 576
Glu His Gly Asp Ser Gln Met Val Ala Trp Ser His Val Thr Val Gly
180 185 190
gga aag ccc atc ctg cag atc ctc gaa gaa cag aaa gag cgt ttc ggc 624
Gly Lys Pro Ile Leu Gln Ile Leu Glu Glu Gln Lys Glu Arg Phe Gly
195 200 205
gag att gac ctg gac gag atc gtg gaa aag act gcc aag gcc ggt tgg 672
Glu Ile Asp Leu Asp Glu Ile Val Glu Lys Thr Ala Lys Ala Gly Trp
210 215 220
gag atc tat aag cgc aag ggg aca acg tac tat ggc atc ggc aac tcc 720
Glu Ile Tyr Lys Arg Lys Gly Thr Thr Tyr Tyr Gly Ile Gly Asn Ser
225 230 235 240
ctg gcc tat atc gcg tcg tct atc ttc aac gac gac cat cgc gta atc 768
Leu Ala Tyr Ile Ala Ser Ser Ile Phe Asn Asp Asp His Arg Val Ile
245 250 255
gcg gtg agc gcc atc ctg gac ggc gag tac ggt gaa tac gat att tgc 816
Ala Val Ser Ala Ile Leu Asp Gly Glu Tyr Gly Glu Tyr Asp Ile Cys
260 265 270
acc ggc gtg ccg gcc att atc acc cgg gat gga ata cgc gaa gtc gtg 864
Thr Gly Val Pro Ala Ile Ile Thr Arg Asp Gly Ile Arg Glu Val Val
275 280 285
gag ttg aac ctg acg gaa gat gaa gag tcc cgg ttc gcg aag tcc aat 912
Glu Leu Asn Leu Thr Glu Asp Glu Glu Ser Arg Phe Ala Lys Ser Asn
290 295 300
gac atc ctg cgc gac tat atg aaa acc atc ggc tac taa 951
Asp Ile Leu Arg Asp Tyr Met Lys Thr Ile Gly Tyr
305 310 315
<210> 40
<211> 316
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 40
Met Lys Arg His Thr Arg Lys Ile Ala Ile Ile Gly Thr Gly Leu Val
1 5 10 15
Gly Ser Ser Cys Ala Tyr Ser Ile Val Asn Gln Gly Ile Cys Glu Glu
20 25 30
Leu Leu Leu Ile Asp Ile Asn His Glu Arg Ala Val Gly Glu Ala Met
35 40 45
Asp Leu Ser His Cys Ile Asn Phe Thr Asn Thr Arg Thr Lys Val Tyr
50 55 60
Ala Gly Ser Tyr Glu Asp Cys Lys Asp Met Asp Ile Val Ile Ile Thr
65 70 75 80
Ala Gly Pro Ala Pro Lys Pro Gly Gln Ser Arg Leu Asp Thr Leu Gly
85 90 95
Ala Ser Ala Lys Ile Met Glu Ser Val Val Gly Gly Val Met Glu Ser
100 105 110
Gly Phe Asp Gly Ile Phe Leu Leu Ala Ser Asn Pro Val Asp Ile Ile
115 120 125
Thr Tyr Gln Val Trp Lys Leu Ser Gly Leu Pro Arg Asn Arg Val Ile
130 135 140
Gly Thr Gly Thr Ser Leu Asp Ser Ser Arg Leu Arg Thr Ile Leu Ser
145 150 155 160
Glu Met Leu His Val Asp Pro Arg Ser Ile His Gly Tyr Ser Leu Gly
165 170 175
Glu His Gly Asp Ser Gln Met Val Ala Trp Ser His Val Thr Val Gly
180 185 190
Gly Lys Pro Ile Leu Gln Ile Leu Glu Glu Gln Lys Glu Arg Phe Gly
195 200 205
Glu Ile Asp Leu Asp Glu Ile Val Glu Lys Thr Ala Lys Ala Gly Trp
210 215 220
Glu Ile Tyr Lys Arg Lys Gly Thr Thr Tyr Tyr Gly Ile Gly Asn Ser
225 230 235 240
Leu Ala Tyr Ile Ala Ser Ser Ile Phe Asn Asp Asp His Arg Val Ile
245 250 255
Ala Val Ser Ala Ile Leu Asp Gly Glu Tyr Gly Glu Tyr Asp Ile Cys
260 265 270
Thr Gly Val Pro Ala Ile Ile Thr Arg Asp Gly Ile Arg Glu Val Val
275 280 285
Glu Leu Asn Leu Thr Glu Asp Glu Glu Ser Arg Phe Ala Lys Ser Asn
290 295 300
Asp Ile Leu Arg Asp Tyr Met Lys Thr Ile Gly Tyr
305 310 315
<210> 41
<211> 954
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Geobacillus stearothermophilus)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(954)
<400> 41
atg aag aat aat ggt ggc gca agg gta gtg gta atc gga gcg ggg ttt 48
Met Lys Asn Asn Gly Gly Ala Arg Val Val Val Ile Gly Ala Gly Phe
1 5 10 15
gtg gga gcg tct tat gtg ttc gcg ctc atg aac cag ggc atc gcc gac 96
Val Gly Ala Ser Tyr Val Phe Ala Leu Met Asn Gln Gly Ile Ala Asp
20 25 30
gag atc gtg ctc atc gac gcc aac gag agc aag gct atc ggc gat gcg 144
Glu Ile Val Leu Ile Asp Ala Asn Glu Ser Lys Ala Ile Gly Asp Ala
35 40 45
atg gac ttc aat cat ggc aag gtc ttt gcg ccc aaa ccg gtg gat atc 192
Met Asp Phe Asn His Gly Lys Val Phe Ala Pro Lys Pro Val Asp Ile
50 55 60
tgg cac ggt gac tac gat gac tgc cgg gac gcc gac ctg gtt gtc atc 240
Trp His Gly Asp Tyr Asp Asp Cys Arg Asp Ala Asp Leu Val Val Ile
65 70 75 80
tgc gct ggc gct aac caa aag ccc ggg gaa acc cgg ctg gac ttg gtc 288
Cys Ala Gly Ala Asn Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val
85 90 95
gat aag aac atc gcg att ttc cga agc atc gtg gag tcc gtc atg gcg 336
Asp Lys Asn Ile Ala Ile Phe Arg Ser Ile Val Glu Ser Val Met Ala
100 105 110
tcg ggc ttc cag ggc ctg ttc ctg gtg gcc aca aac ccg gtc gac ata 384
Ser Gly Phe Gln Gly Leu Phe Leu Val Ala Thr Asn Pro Val Asp Ile
115 120 125
ctc acg tat gcc acc tgg aag ttc tcc gga ctg ccg cac gaa cgg gtc 432
Leu Thr Tyr Ala Thr Trp Lys Phe Ser Gly Leu Pro His Glu Arg Val
130 135 140
atc ggg agc ggt acg att ctg gac acg gca cgc ttc cgc ttc ctg ttg 480
Ile Gly Ser Gly Thr Ile Leu Asp Thr Ala Arg Phe Arg Phe Leu Leu
145 150 155 160
ggc gag tac ttc tcg gtc gcc cct cag aac gtc cat gcc tac atc atc 528
Gly Glu Tyr Phe Ser Val Ala Pro Gln Asn Val His Ala Tyr Ile Ile
165 170 175
ggc gag cac ggc gat acc gaa ctg cca gtg tgg tcg cag gcc tat atc 576
Gly Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser Gln Ala Tyr Ile
180 185 190
ggc gtc atg ccc atc cgc aaa ctg gtg gag tcg aag ggc gaa gaa gcc 624
Gly Val Met Pro Ile Arg Lys Leu Val Glu Ser Lys Gly Glu Glu Ala
195 200 205
cag aaa gac ctg gaa cgc atc ttc gtc aat gtg cgc gat gca gcg tac 672
Gln Lys Asp Leu Glu Arg Ile Phe Val Asn Val Arg Asp Ala Ala Tyr
210 215 220
cag atc att gaa aag aaa ggg gcc acc tat tac ggg atc gcg atg ggc 720
Gln Ile Ile Glu Lys Lys Gly Ala Thr Tyr Tyr Gly Ile Ala Met Gly
225 230 235 240
ctc gcc cgt gtc act cgg gcg atc ctc cac aac gag aac gcc atc ttg 768
Leu Ala Arg Val Thr Arg Ala Ile Leu His Asn Glu Asn Ala Ile Leu
245 250 255
acc gtc agc gcc tat ctg gac ggc ctt tac ggc gaa cgc gac gtg tac 816
Thr Val Ser Ala Tyr Leu Asp Gly Leu Tyr Gly Glu Arg Asp Val Tyr
260 265 270
atc ggc gtt ccg gcc gtg atc aat cgg aac ggt atc cgc gag gtg atc 864
Ile Gly Val Pro Ala Val Ile Asn Arg Asn Gly Ile Arg Glu Val Ile
275 280 285
gaa atc gag ctc aac gat gac gag aaa aac cgg ttc cat cac agt gcg 912
Glu Ile Glu Leu Asn Asp Asp Glu Lys Asn Arg Phe His His Ser Ala
290 295 300
gcc acc ctg aag tcc gtc ctg gcc cgt gcc ttc acc cgc taa 954
Ala Thr Leu Lys Ser Val Leu Ala Arg Ala Phe Thr Arg
305 310 315
<210> 42
<211> 317
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 42
Met Lys Asn Asn Gly Gly Ala Arg Val Val Val Ile Gly Ala Gly Phe
1 5 10 15
Val Gly Ala Ser Tyr Val Phe Ala Leu Met Asn Gln Gly Ile Ala Asp
20 25 30
Glu Ile Val Leu Ile Asp Ala Asn Glu Ser Lys Ala Ile Gly Asp Ala
35 40 45
Met Asp Phe Asn His Gly Lys Val Phe Ala Pro Lys Pro Val Asp Ile
50 55 60
Trp His Gly Asp Tyr Asp Asp Cys Arg Asp Ala Asp Leu Val Val Ile
65 70 75 80
Cys Ala Gly Ala Asn Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val
85 90 95
Asp Lys Asn Ile Ala Ile Phe Arg Ser Ile Val Glu Ser Val Met Ala
100 105 110
Ser Gly Phe Gln Gly Leu Phe Leu Val Ala Thr Asn Pro Val Asp Ile
115 120 125
Leu Thr Tyr Ala Thr Trp Lys Phe Ser Gly Leu Pro His Glu Arg Val
130 135 140
Ile Gly Ser Gly Thr Ile Leu Asp Thr Ala Arg Phe Arg Phe Leu Leu
145 150 155 160
Gly Glu Tyr Phe Ser Val Ala Pro Gln Asn Val His Ala Tyr Ile Ile
165 170 175
Gly Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser Gln Ala Tyr Ile
180 185 190
Gly Val Met Pro Ile Arg Lys Leu Val Glu Ser Lys Gly Glu Glu Ala
195 200 205
Gln Lys Asp Leu Glu Arg Ile Phe Val Asn Val Arg Asp Ala Ala Tyr
210 215 220
Gln Ile Ile Glu Lys Lys Gly Ala Thr Tyr Tyr Gly Ile Ala Met Gly
225 230 235 240
Leu Ala Arg Val Thr Arg Ala Ile Leu His Asn Glu Asn Ala Ile Leu
245 250 255
Thr Val Ser Ala Tyr Leu Asp Gly Leu Tyr Gly Glu Arg Asp Val Tyr
260 265 270
Ile Gly Val Pro Ala Val Ile Asn Arg Asn Gly Ile Arg Glu Val Ile
275 280 285
Glu Ile Glu Leu Asn Asp Asp Glu Lys Asn Arg Phe His His Ser Ala
290 295 300
Ala Thr Leu Lys Ser Val Leu Ala Arg Ala Phe Thr Arg
305 310 315
<210> 43
<211> 915
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Deinococcus radiodurans)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(915)
<400> 43
atg aaa gtc ggg gta gtc ggt acg gga ttt gta ggt tca aca gca gcg 48
Met Lys Val Gly Val Val Gly Thr Gly Phe Val Gly Ser Thr Ala Ala
1 5 10 15
ttc gct ctt gtt ctc agg ggc agc tgc agc gaa ctg gtg ctc gtc gac 96
Phe Ala Leu Val Leu Arg Gly Ser Cys Ser Glu Leu Val Leu Val Asp
20 25 30
cgc gat gaa gat cgg gcc caa gcg gaa gcc gag gac atc gcg cac gcc 144
Arg Asp Glu Asp Arg Ala Gln Ala Glu Ala Glu Asp Ile Ala His Ala
35 40 45
gcc ccg gtg agt cac ggc acc cgc gtg tgg cat ggc ggc cac tcg gaa 192
Ala Pro Val Ser His Gly Thr Arg Val Trp His Gly Gly His Ser Glu
50 55 60
ctc gcg gac gcc cag gtc gtg att ctg acc gca ggc gcc aac cag aaa 240
Leu Ala Asp Ala Gln Val Val Ile Leu Thr Ala Gly Ala Asn Gln Lys
65 70 75 80
ccg ggc gag agc cgg ctg gac ctg ttg gag aag aac gcg gac atc ttc 288
Pro Gly Glu Ser Arg Leu Asp Leu Leu Glu Lys Asn Ala Asp Ile Phe
85 90 95
cgc gag ctc gtc ccc cag atc acc cgt gcg gct ccc gac gcg gtc ttg 336
Arg Glu Leu Val Pro Gln Ile Thr Arg Ala Ala Pro Asp Ala Val Leu
100 105 110
ctc gtg acc agc aac cct gtc gat ctg ttg acc gac ctg gcc act cag 384
Leu Val Thr Ser Asn Pro Val Asp Leu Leu Thr Asp Leu Ala Thr Gln
115 120 125
ctg gca ccc ggc cag ccg gtg ata ggc tcc ggc acg gtc ctc gac tct 432
Leu Ala Pro Gly Gln Pro Val Ile Gly Ser Gly Thr Val Leu Asp Ser
130 135 140
gcc cgg ttc cga cat ctg atg gcc caa cac gcc ggg gtc gac gga acc 480
Ala Arg Phe Arg His Leu Met Ala Gln His Ala Gly Val Asp Gly Thr
145 150 155 160
cat gca cac ggt tat gtg ctg ggc gag cac ggc gat tcc gaa gtg ctg 528
His Ala His Gly Tyr Val Leu Gly Glu His Gly Asp Ser Glu Val Leu
165 170 175
gcc tgg tcg tcc gcc atg gtg gcc ggc atg cca gtg gcg gac ttc atg 576
Ala Trp Ser Ser Ala Met Val Ala Gly Met Pro Val Ala Asp Phe Met
180 185 190
cag gcc cag aac ctc ccg tgg aat gaa cag gtt cgc gcg aag atc gac 624
Gln Ala Gln Asn Leu Pro Trp Asn Glu Gln Val Arg Ala Lys Ile Asp
195 200 205
gaa ggc acc cgt aat gcg gcc gcg tcg atc atc gag ggc aaa cgc gcc 672
Glu Gly Thr Arg Asn Ala Ala Ala Ser Ile Ile Glu Gly Lys Arg Ala
210 215 220
acc tat tac ggc atc ggt gcc gcg ctg gcc cgg atc acc gag gcc gtc 720
Thr Tyr Tyr Gly Ile Gly Ala Ala Leu Ala Arg Ile Thr Glu Ala Val
225 230 235 240
ctg cgc gat cgc cgt gcc gtg ctg acc gtc agc gct ccg acg ccg gag 768
Leu Arg Asp Arg Arg Ala Val Leu Thr Val Ser Ala Pro Thr Pro Glu
245 250 255
tac gga gtg agc ctg tcc ctt ccc cgg gtg gtc gga cgc cag ggc gtc 816
Tyr Gly Val Ser Leu Ser Leu Pro Arg Val Val Gly Arg Gln Gly Val
260 265 270
ttg tcg acg ctc cat ccg aag ctg acg ggg gat gag cag cag aag ctg 864
Leu Ser Thr Leu His Pro Lys Leu Thr Gly Asp Glu Gln Gln Lys Leu
275 280 285
gaa cag tcg gcg ggt gtc ctg cgg ggg ttc aag caa cag ctg ggg ctg 912
Glu Gln Ser Ala Gly Val Leu Arg Gly Phe Lys Gln Gln Leu Gly Leu
290 295 300
taa 915
<210> 44
<211> 304
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 44
Met Lys Val Gly Val Val Gly Thr Gly Phe Val Gly Ser Thr Ala Ala
1 5 10 15
Phe Ala Leu Val Leu Arg Gly Ser Cys Ser Glu Leu Val Leu Val Asp
20 25 30
Arg Asp Glu Asp Arg Ala Gln Ala Glu Ala Glu Asp Ile Ala His Ala
35 40 45
Ala Pro Val Ser His Gly Thr Arg Val Trp His Gly Gly His Ser Glu
50 55 60
Leu Ala Asp Ala Gln Val Val Ile Leu Thr Ala Gly Ala Asn Gln Lys
65 70 75 80
Pro Gly Glu Ser Arg Leu Asp Leu Leu Glu Lys Asn Ala Asp Ile Phe
85 90 95
Arg Glu Leu Val Pro Gln Ile Thr Arg Ala Ala Pro Asp Ala Val Leu
100 105 110
Leu Val Thr Ser Asn Pro Val Asp Leu Leu Thr Asp Leu Ala Thr Gln
115 120 125
Leu Ala Pro Gly Gln Pro Val Ile Gly Ser Gly Thr Val Leu Asp Ser
130 135 140
Ala Arg Phe Arg His Leu Met Ala Gln His Ala Gly Val Asp Gly Thr
145 150 155 160
His Ala His Gly Tyr Val Leu Gly Glu His Gly Asp Ser Glu Val Leu
165 170 175
Ala Trp Ser Ser Ala Met Val Ala Gly Met Pro Val Ala Asp Phe Met
180 185 190
Gln Ala Gln Asn Leu Pro Trp Asn Glu Gln Val Arg Ala Lys Ile Asp
195 200 205
Glu Gly Thr Arg Asn Ala Ala Ala Ser Ile Ile Glu Gly Lys Arg Ala
210 215 220
Thr Tyr Tyr Gly Ile Gly Ala Ala Leu Ala Arg Ile Thr Glu Ala Val
225 230 235 240
Leu Arg Asp Arg Arg Ala Val Leu Thr Val Ser Ala Pro Thr Pro Glu
245 250 255
Tyr Gly Val Ser Leu Ser Leu Pro Arg Val Val Gly Arg Gln Gly Val
260 265 270
Leu Ser Thr Leu His Pro Lys Leu Thr Gly Asp Glu Gln Gln Lys Leu
275 280 285
Glu Gln Ser Ala Gly Val Leu Arg Gly Phe Lys Gln Gln Leu Gly Leu
290 295 300
<210> 45
<211> 948
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (вариант ЛДГ Plasmodium ovale)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(948)
<400> 45
atg gca cct aaa ccc aaa att gta cta gtc gga agc ggc atg atc ggc 48
Met Ala Pro Lys Pro Lys Ile Val Leu Val Gly Ser Gly Met Ile Gly
1 5 10 15
gga gta atg gca acc ctc atc gtc cag aag aat ctg ggc gat gtc gtc 96
Gly Val Met Ala Thr Leu Ile Val Gln Lys Asn Leu Gly Asp Val Val
20 25 30
atg ttc gat atc gtc aag aac atg ccc ctc ggc aag gcc ctg gac acc 144
Met Phe Asp Ile Val Lys Asn Met Pro Leu Gly Lys Ala Leu Asp Thr
35 40 45
tca cat acc aac gtg atg gcc tac tcc aac tgc cag gtg acc ggc agc 192
Ser His Thr Asn Val Met Ala Tyr Ser Asn Cys Gln Val Thr Gly Ser
50 55 60
aat acc tac gag gac ttg aag ggt gcg gac gtg gtg gtg aca gcc ggc 240
Asn Thr Tyr Glu Asp Leu Lys Gly Ala Asp Val Val Val Thr Ala Gly
65 70 75 80
ttc acg aag gct ccg ggc aag tcc gac aag gaa tgg aac cgc gac gat 288
Phe Thr Lys Ala Pro Gly Lys Ser Asp Lys Glu Trp Asn Arg Asp Asp
85 90 95
ctg ctc ccg ctg aat aac aag att atg atc gag atc ggc ggt cac atc 336
Leu Leu Pro Leu Asn Asn Lys Ile Met Ile Glu Ile Gly Gly His Ile
100 105 110
aag aac tat tgt ccg aac gcg ttc atc atc gtg gtc act aac cca gcc 384
Lys Asn Tyr Cys Pro Asn Ala Phe Ile Ile Val Val Thr Asn Pro Ala
115 120 125
gac gtg atg gtg cag ctg ctg cac cag cat agc ggc gtg agc aag aac 432
Asp Val Met Val Gln Leu Leu His Gln His Ser Gly Val Ser Lys Asn
130 135 140
aag atc gtc ggc ctg ggt ggt gtg ctg gat acc tcc cgg ctg aag tat 480
Lys Ile Val Gly Leu Gly Gly Val Leu Asp Thr Ser Arg Leu Lys Tyr
145 150 155 160
tac atc tcg cag aag ctc aat gtc tgc ccc cgt gac gtc aac gcg cac 528
Tyr Ile Ser Gln Lys Leu Asn Val Cys Pro Arg Asp Val Asn Ala His
165 170 175
atc gtc ggg gcg cac ggg aac aaa atg gtc gtt ctg aag cgc tat atc 576
Ile Val Gly Ala His Gly Asn Lys Met Val Val Leu Lys Arg Tyr Ile
180 185 190
acc gtg ggg ggc atc ccg ctg caa gag ttc atc aac aac aaa aag atc 624
Thr Val Gly Gly Ile Pro Leu Gln Glu Phe Ile Asn Asn Lys Lys Ile
195 200 205
acc gac gcc gag ctc gat gcg ata ttc gat cgg acg gtc aat acg gcc 672
Thr Asp Ala Glu Leu Asp Ala Ile Phe Asp Arg Thr Val Asn Thr Ala
210 215 220
ctt gag atc gtc aac tac cac gcc agt ccg tac gtg gct ccg gcc gcg 720
Leu Glu Ile Val Asn Tyr His Ala Ser Pro Tyr Val Ala Pro Ala Ala
225 230 235 240
gcc atc atc gag atg gcc gag tcg tat ttg aaa gac ctg aaa aaa gtg 768
Ala Ile Ile Glu Met Ala Glu Ser Tyr Leu Lys Asp Leu Lys Lys Val
245 250 255
ctg att tgc tcg acc ctg ttg gaa ggc cag tac ggc cat acg ggc gtg 816
Leu Ile Cys Ser Thr Leu Leu Glu Gly Gln Tyr Gly His Thr Gly Val
260 265 270
ttt ggc ggg acg ccc ctg gtc ctc gga tgc aat ggc gtc gaa cag gtt 864
Phe Gly Gly Thr Pro Leu Val Leu Gly Cys Asn Gly Val Glu Gln Val
275 280 285
ttc gaa ctc cag ctg aac gcg gaa gaa aag aaa atg ttc gac gac gcc 912
Phe Glu Leu Gln Leu Asn Ala Glu Glu Lys Lys Met Phe Asp Asp Ala
290 295 300
atc gcc gaa acc aag cgc atg aag gcc ctg gcg taa 948
Ile Ala Glu Thr Lys Arg Met Lys Ala Leu Ala
305 310 315
<210> 46
<211> 315
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 46
Met Ala Pro Lys Pro Lys Ile Val Leu Val Gly Ser Gly Met Ile Gly
1 5 10 15
Gly Val Met Ala Thr Leu Ile Val Gln Lys Asn Leu Gly Asp Val Val
20 25 30
Met Phe Asp Ile Val Lys Asn Met Pro Leu Gly Lys Ala Leu Asp Thr
35 40 45
Ser His Thr Asn Val Met Ala Tyr Ser Asn Cys Gln Val Thr Gly Ser
50 55 60
Asn Thr Tyr Glu Asp Leu Lys Gly Ala Asp Val Val Val Thr Ala Gly
65 70 75 80
Phe Thr Lys Ala Pro Gly Lys Ser Asp Lys Glu Trp Asn Arg Asp Asp
85 90 95
Leu Leu Pro Leu Asn Asn Lys Ile Met Ile Glu Ile Gly Gly His Ile
100 105 110
Lys Asn Tyr Cys Pro Asn Ala Phe Ile Ile Val Val Thr Asn Pro Ala
115 120 125
Asp Val Met Val Gln Leu Leu His Gln His Ser Gly Val Ser Lys Asn
130 135 140
Lys Ile Val Gly Leu Gly Gly Val Leu Asp Thr Ser Arg Leu Lys Tyr
145 150 155 160
Tyr Ile Ser Gln Lys Leu Asn Val Cys Pro Arg Asp Val Asn Ala His
165 170 175
Ile Val Gly Ala His Gly Asn Lys Met Val Val Leu Lys Arg Tyr Ile
180 185 190
Thr Val Gly Gly Ile Pro Leu Gln Glu Phe Ile Asn Asn Lys Lys Ile
195 200 205
Thr Asp Ala Glu Leu Asp Ala Ile Phe Asp Arg Thr Val Asn Thr Ala
210 215 220
Leu Glu Ile Val Asn Tyr His Ala Ser Pro Tyr Val Ala Pro Ala Ala
225 230 235 240
Ala Ile Ile Glu Met Ala Glu Ser Tyr Leu Lys Asp Leu Lys Lys Val
245 250 255
Leu Ile Cys Ser Thr Leu Leu Glu Gly Gln Tyr Gly His Thr Gly Val
260 265 270
Phe Gly Gly Thr Pro Leu Val Leu Gly Cys Asn Gly Val Glu Gln Val
275 280 285
Phe Glu Leu Gln Leu Asn Ala Glu Glu Lys Lys Met Phe Asp Asp Ala
290 295 300
Ile Ala Glu Thr Lys Arg Met Lys Ala Leu Ala
305 310 315
<210> 47
<211> 933
<212> DNA/ДНК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция (ЛДГ Thermus thermophilus)
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(933)
<400> 47
atg aaa gta gga ata gtc ggc tct gga atg gtt gga tcg gca acg gca 48
Met Lys Val Gly Ile Val Gly Ser Gly Met Val Gly Ser Ala Thr Ala
1 5 10 15
tac gca ctc gca ctc ctc ggc gtg gcg cgg gaa gtc gtg ctg gtc gat 96
Tyr Ala Leu Ala Leu Leu Gly Val Ala Arg Glu Val Val Leu Val Asp
20 25 30
ctc gac cgc aag ctg gcg cag gcc cat gcc gag gat atc ctg cac gcg 144
Leu Asp Arg Lys Leu Ala Gln Ala His Ala Glu Asp Ile Leu His Ala
35 40 45
acc ccg ttc gcg cat ccc gtg tgg gtc cgt gcc ggc agc tat ggc gac 192
Thr Pro Phe Ala His Pro Val Trp Val Arg Ala Gly Ser Tyr Gly Asp
50 55 60
ctg gaa ggg gcg aga gcg gtc gtc ctg gcg gcc ggg gtg gcc cag cgg 240
Leu Glu Gly Ala Arg Ala Val Val Leu Ala Ala Gly Val Ala Gln Arg
65 70 75 80
cca ggc gaa acc cgc ctc cag ctg ctc gat cgc aac gcc cag gtg ttt 288
Pro Gly Glu Thr Arg Leu Gln Leu Leu Asp Arg Asn Ala Gln Val Phe
85 90 95
gcc caa gtg gta ccg cgg gtt ctg gaa gct gcc ccc gaa gcc gtg ttg 336
Ala Gln Val Val Pro Arg Val Leu Glu Ala Ala Pro Glu Ala Val Leu
100 105 110
ctg gtc gcc acc aat ccc gtc gac gtg atg acc cag gtc gcc tac cgc 384
Leu Val Ala Thr Asn Pro Val Asp Val Met Thr Gln Val Ala Tyr Arg
115 120 125
ctg agc ggt ctg ccg ccg gga agg gtc gtg ggg agc ggc acg atc ctg 432
Leu Ser Gly Leu Pro Pro Gly Arg Val Val Gly Ser Gly Thr Ile Leu
130 135 140
gac aca gcg cgg ttc cgg gcc ttg ctt gcc gag tat ctg cgg gtg gct 480
Asp Thr Ala Arg Phe Arg Ala Leu Leu Ala Glu Tyr Leu Arg Val Ala
145 150 155 160
ccg cag tcg gtc cac gcg tac gtg ctc ggc gaa cac ggc gac tcg gaa 528
Pro Gln Ser Val His Ala Tyr Val Leu Gly Glu His Gly Asp Ser Glu
165 170 175
gtc ctc gtg tgg agc tcc gcg caa gtc ggg ggt gtg ccg ctt ctg gag 576
Val Leu Val Trp Ser Ser Ala Gln Val Gly Gly Val Pro Leu Leu Glu
180 185 190
ttc gcg gaa gcg cgt ggg cgt gcc cta tcg cct gag gac agg gct cgg 624
Phe Ala Glu Ala Arg Gly Arg Ala Leu Ser Pro Glu Asp Arg Ala Arg
195 200 205
atc gac gag ggc gtg cga cgg gcc gcc tac cgc atc atc gag ggc aag 672
Ile Asp Glu Gly Val Arg Arg Ala Ala Tyr Arg Ile Ile Glu Gly Lys
210 215 220
ggc gcg act tat tac ggc atc ggt gcc ggc ctg gcg cgt ctg gtc cgc 720
Gly Ala Thr Tyr Tyr Gly Ile Gly Ala Gly Leu Ala Arg Leu Val Arg
225 230 235 240
gcg atc ttg acc gac gag aaa ggc gtc tat acg gtg tcc gcc ttc acc 768
Ala Ile Leu Thr Asp Glu Lys Gly Val Tyr Thr Val Ser Ala Phe Thr
245 250 255
ccc gag gtt gag ggt gtg ctg gaa gtc agt ctg tcc ttg ccg cgc att 816
Pro Glu Val Glu Gly Val Leu Glu Val Ser Leu Ser Leu Pro Arg Ile
260 265 270
ctc ggc gcc ggc ggc gtc gag ggc acc gtg tac ccg tcc ctg tca ccc 864
Leu Gly Ala Gly Gly Val Glu Gly Thr Val Tyr Pro Ser Leu Ser Pro
275 280 285
gag gaa cgc gag gcc ctg cgc cgc agc gcc gaa atc ctc aag gaa gcg 912
Glu Glu Arg Glu Ala Leu Arg Arg Ser Ala Glu Ile Leu Lys Glu Ala
290 295 300
gcg ttc gcc ctg ggt ttc taa 933
Ala Phe Ala Leu Gly Phe
305 310
<210> 48
<211> 310
<212> PRT/БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетическая конструкция
<400> 48
Met Lys Val Gly Ile Val Gly Ser Gly Met Val Gly Ser Ala Thr Ala
1 5 10 15
Tyr Ala Leu Ala Leu Leu Gly Val Ala Arg Glu Val Val Leu Val Asp
20 25 30
Leu Asp Arg Lys Leu Ala Gln Ala His Ala Glu Asp Ile Leu His Ala
35 40 45
Thr Pro Phe Ala His Pro Val Trp Val Arg Ala Gly Ser Tyr Gly Asp
50 55 60
Leu Glu Gly Ala Arg Ala Val Val Leu Ala Ala Gly Val Ala Gln Arg
65 70 75 80
Pro Gly Glu Thr Arg Leu Gln Leu Leu Asp Arg Asn Ala Gln Val Phe
85 90 95
Ala Gln Val Val Pro Arg Val Leu Glu Ala Ala Pro Glu Ala Val Leu
100 105 110
Leu Val Ala Thr Asn Pro Val Asp Val Met Thr Gln Val Ala Tyr Arg
115 120 125
Leu Ser Gly Leu Pro Pro Gly Arg Val Val Gly Ser Gly Thr Ile Leu
130 135 140
Asp Thr Ala Arg Phe Arg Ala Leu Leu Ala Glu Tyr Leu Arg Val Ala
145 150 155 160
Pro Gln Ser Val His Ala Tyr Val Leu Gly Glu His Gly Asp Ser Glu
165 170 175
Val Leu Val Trp Ser Ser Ala Gln Val Gly Gly Val Pro Leu Leu Glu
180 185 190
Phe Ala Glu Ala Arg Gly Arg Ala Leu Ser Pro Glu Asp Arg Ala Arg
195 200 205
Ile Asp Glu Gly Val Arg Arg Ala Ala Tyr Arg Ile Ile Glu Gly Lys
210 215 220
Gly Ala Thr Tyr Tyr Gly Ile Gly Ala Gly Leu Ala Arg Leu Val Arg
225 230 235 240
Ala Ile Leu Thr Asp Glu Lys Gly Val Tyr Thr Val Ser Ala Phe Thr
245 250 255
Pro Glu Val Glu Gly Val Leu Glu Val Ser Leu Ser Leu Pro Arg Ile
260 265 270
Leu Gly Ala Gly Gly Val Glu Gly Thr Val Tyr Pro Ser Leu Ser Pro
275 280 285
Glu Glu Arg Glu Ala Leu Arg Arg Ser Ala Glu Ile Leu Lys Glu Ala
290 295 300
Ala Phe Ala Leu Gly Phe
305 310
<210> 49
<211> 229
<212> DNA/ДНК
<213> Промотор МДГ Methanococcus capsulatus Bath
<400> 49
tttgcctcga tcggcggtcc ttgtgacagg gagatattcc cgacggatcc ggggcattcg 60
agcggaaccg cccgccgtgg gagtttttcc agcgagcatt cgagagtttt tcaaggcggc 120
ttcgaggggt tattccgtaa cgccgccgac atgatctgtc ccagaatctc cgccgctgtt 180
cgtagagcgc cgatgcaggg tcggcatcaa tcattcttgg aggagacac 229
<210> 50
<211> 114
<212> DNA/ДНК
<213> Область промотора sgaMethylosinus trichosporium OB3b
<400> 50
ttttttgaga ccacgcggcc cgacgcgcaa ggaagctttc gggcgcgcgc gcgtccggcg 60
aagacaactc gaaccgatga gctcgaaggg agccctagac aggaggtaaa aatg 114
<210> 51
<211> 707
<212> DNA/ДНК
<213> Промотор maxF Methylmonas sp. 16a
<400> 51
gcgccgtatg ctttcgaatc cgccaagacc gggcatggat aaatccatga ccaccacatc 60
gggcaattgc tttgaataga gttggcaagc ggtctcgcca cgatcggctt catagatctc 120
gccgatacga tccgacagcg atagataggt cttgtagccg gtacgaacca cggcgtgatc 180
atctaccaat aaaacgctga ttttactcgc cactggaaaa tttcctcctc aggtcgtcaa 240
gggataaaga tatgggacaa gtccagtctg atgccaggcg gacttggtgt gccttttttt 300
atgatgacgc tttatccgtg cttaaaccat gggagctttt cccgtttcca atttcgatcc 360
ttggcgagat aggaatattt ccgtgcatga ttgcgtcgat ttcacatcga tttcatggat 420
tgttccgtaa cgttagccag cccggcttct ataacatttg cgccagcgtg gcctggtggt 480
cggtaacccg tgatgcggtt atgatcaaca aagctggttt tcaacgacta attctgatct 540
tcaggtcgcg cctcacttat agcgataaaa atcctggagg aaacatgcaa caactcgatt 600
tgcgcatagt cgggaaaacc gcggccttgt tggctggtgg ccttctgagc gtggcgcaac 660
ccgcatcggc gaacaaggag ctggaacagc tcggaggatt taaaatg 707
<210> 52
<211> 281
<212> DNA/ДНК
<213> Промотор hps Methylmonas sp. 16a
<400> 52
cagatagtaa gcgctaagga ttggggtgcg tcgccggtcg cggcggcgct cctcgacggc 60
agagttggtg ccaggttggc ggatgattga tgccgaatat tacgcgacca attctcgagg 120
caaatgaact gtgagctact gagttgcagg cattgacagc catcccattt ctatcataca 180
gttacggacg catcacgagt aggtgataag cctagcagat tgcggcagtt ggcaaaatca 240
gctattacta ataattaaaa actttcggag cacatcaaat g 281
<210> 53
<211> 317
<212> PRT/БЕЛОК
<213> Staphylococcus simiae
<400> 53
Met Asn Gln Phe Lys Gly Asn Lys Val Val Leu Ile Gly Asn Gly Ala
1 5 10 15
Val Gly Ser Ser Tyr Ala Phe Ser Leu Val Asn Gln Ser Ile Val Asp
20 25 30
Glu Leu Val Ile Ile Asp Leu Asp Thr Glu Lys Val Lys Gly Asp Val
35 40 45
Met Asp Leu Lys His Ala Thr Pro Tyr Ser Pro Thr Thr Val Arg Val
50 55 60
Lys Ala Gly Glu Tyr Ser Asp Cys His Asp Ala Asp Leu Val Val Ile
65 70 75 80
Cys Ala Gly Ala Ala Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val
85 90 95
Ser Lys Asn Leu Lys Ile Phe Lys Ser Ile Val Asp Glu Val Met Ala
100 105 110
Ser Gln Phe Asp Gly Ile Phe Leu Val Ala Thr Asn Pro Val Asp Ile
115 120 125
Leu Ser Tyr Ala Thr Trp Lys Phe Ser Gly Leu Pro Lys Glu Arg Val
130 135 140
Ile Gly Ser Gly Thr Ile Leu Asp Ser Ala Arg Phe Arg Leu Leu Leu
145 150 155 160
Ser Glu Glu Phe Asp Val Ala Pro Ser Ser Val His Ala Gln Ile Ile
165 170 175
Gly Glu His Gly Asp Thr Glu Leu Ala Val Trp Ser His Ala Asn Ile
180 185 190
Ala Gly Gln Pro Leu Lys Glu Ile Leu Glu Lys Arg Pro Gln Glu Lys
195 200 205
Glu Lys Val Glu Gln Ile Phe Val Gln Thr Arg Asp Ala Ala Tyr Asp
210 215 220
Ile Ile Lys Ala Lys Gly Ala Thr Tyr Tyr Gly Val Ala Met Gly Leu
225 230 235 240
Thr Arg Ile Thr Glu Ala Ile Phe Arg Asn Glu Asp Ala Val Leu Thr
245 250 255
Ile Ser Ala Leu Leu Glu Gly Glu Tyr Gly Gln Glu Asp Leu Tyr Ile
260 265 270
Gly Val Pro Ala Val Ile Asn Arg Gly Gly Ile Arg Asn Val Val Glu
275 280 285
Thr Pro Leu Asn Glu Glu Glu Arg Thr Gln Phe Ala His Ser Ala His
290 295 300
Thr Leu Lys Asp Ile Met Ser Lys Ala Asp Glu Leu Lys
305 310 315
<210> 54
<211> 317
<212> PRT/БЕЛОК
<213> Staphylococcus vitulinus
<400> 54
Met Lys Gln Phe Lys Gly Lys Lys Val Val Leu Val Gly Asn Gly Ala
1 5 10 15
Val Gly Ser Ser Tyr Ala Phe Ser Met Ile Asn Gln Gly Ile Cys Asp
20 25 30
Glu Phe Val Ile Ile Asp Leu Asp Lys Lys Lys Val Asn Gly Asp Val
35 40 45
Met Asp Leu Asn His Gly Thr Val Tyr Gly Pro Ser Pro Ile Lys Val
50 55 60
Lys Ala Gly Glu Tyr Lys Asp Cys Asn Asp Ala Asp Leu Val Val Ile
65 70 75 80
Cys Ala Gly Ala Ala Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val
85 90 95
Ala Lys Asn Met Lys Ile Phe Lys Ser Ile Val Asp Glu Ile Met Arg
100 105 110
Ser Gly Phe Asp Gly Ile Phe Leu Ile Ala Thr Asn Pro Val Asp Val
115 120 125
Leu Thr Tyr Ala Thr Met Lys Phe Ser Gly Leu Pro Lys Glu Arg Val
130 135 140
Ile Gly Ser Gly Thr Ile Leu Asp Thr Ala Arg Phe Arg Tyr Leu Leu
145 150 155 160
Ser Glu Glu Phe Asp Val Ala Pro Gln Ser Val His Ala Asn Ile Ile
165 170 175
Gly Glu His Gly Asp Ser Glu Leu Pro Val Trp Ser His Ala Asn Ile
180 185 190
Ala Gly Lys Pro Leu Arg Ser Ile Ile Glu Gln Asp Asp Ser Arg Lys
195 200 205
His Arg Val Glu Glu Ile Phe Val Gln Thr Arg Asp Ala Ala Tyr Glu
210 215 220
Ile Ile Glu Ala Lys Gly Ala Thr Tyr Tyr Gly Val Ala Met Gly Leu
225 230 235 240
Met Arg Ile Thr Lys Ala Ile Leu Asn Asn Gln Asp Val Val Leu Thr
245 250 255
Val Ser Ala Tyr Leu Glu Gly Glu Tyr Gly His Glu Gly Val Tyr Ile
260 265 270
Gly Val Pro Ala Leu Ile Asn Arg Ser Gly Ile Lys Glu Val Ile Glu
275 280 285
Ile Pro Leu Asp Glu Glu Glu Thr Ser Leu Phe Asn His Ser Val Lys
290 295 300
Val Leu Lys Asp Ile Gln Asp Pro Phe Glu Lys Glu Phe
305 310 315
<210> 55
<211> 318
<212> PRT/БЕЛОК
<213> Staphylococcus lentus
<400> 55
Met Lys Gln Phe Lys Gly Asn Lys Val Val Leu Val Gly Asn Gly Phe
1 5 10 15
Val Gly Ser Ser Tyr Ala Phe Ser Met Ile Asn Gln Gly Ile Cys Asp
20 25 30
Glu Phe Val Ile Ile Asp Leu Asp Lys Lys Lys Val Asn Gly Asp Val
35 40 45
Met Asp Leu Asn His Gly Ser Val Tyr Gly Pro Ser Pro Met Lys Val
50 55 60
Lys Ala Gly Glu Tyr Lys Asp Cys Glu Asp Ala Asp Leu Val Val Ile
65 70 75 80
Cys Ala Gly Ala Ala Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val
85 90 95
Ala Lys Asn Met Lys Ile Phe Lys Asn Ile Val Thr Glu Ile Met Asp
100 105 110
Ser Gly Phe Asp Gly Ile Phe Leu Ile Ala Thr Asn Pro Val Asp Ile
115 120 125
Leu Ser Tyr Ala Thr Met Lys Phe Ser Gly Leu Pro Lys Glu Arg Val
130 135 140
Ile Gly Ser Gly Thr Ile Leu Asp Thr Ala Arg Phe Lys Tyr Leu Leu
145 150 155 160
Ser Glu Glu Phe Asp Val Ala Pro Gln Ser Val His Ala Asn Ile Ile
165 170 175
Gly Glu His Gly Asp Ser Glu Leu Ala Val Trp Ser His Ala Asn Ile
180 185 190
Ala Gly Lys Pro Leu Arg Ser Ile Ile Glu Lys Asp Glu Thr Arg Lys
195 200 205
His Arg Val Glu Glu Ile Phe Val Glu Thr Arg Asp Ala Ala Tyr Glu
210 215 220
Ile Ile Glu Ser Lys Gly Ala Thr Phe Tyr Gly Val Ala Met Gly Leu
225 230 235 240
Met Arg Ile Thr Lys Ala Ile Leu Asn Asn Gln Asp Val Val Leu Thr
245 250 255
Val Ser Ala Tyr Leu Glu Gly Glu Tyr Gly His Glu Asp Val Tyr Ile
260 265 270
Gly Val Pro Ala Leu Val Asn Arg Thr Gly Val Arg Glu Val Ile Glu
275 280 285
Met Pro Leu Asn Asp Glu Glu Thr Arg Leu Phe Asp Asn Ser Val Lys
290 295 300
Val Leu Lys Asp Ile Gln Ala Pro Phe Glu Lys Glu Phe Lys
305 310 315
<210> 56
<211> 315
<212> PRT/БЕЛОК
<213> Macrococcus caseolyticus
<400> 56
Met Glu Lys Phe Lys Gly Asn Lys Val Val Leu Val Gly Asn Gly Ala
1 5 10 15
Val Gly Ser Ser Tyr Ala Phe Ala Met Leu Asn Gln Gly Ala Cys Asp
20 25 30
Glu Phe Val Ile Ile Asp Leu Asn Glu Asp Lys Ala Lys Gly Asp Ala
35 40 45
Met Asp Leu Asn His Gly Val Val Tyr Ala Pro Ser Pro Met Gln Val
50 55 60
Lys Tyr Gly Thr Tyr Glu Asp Cys His Asp Ala Ser Leu Ile Val Ile
65 70 75 80
Cys Ala Gly Ala Ala Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val
85 90 95
Gly Lys Asn Met Lys Ile Phe Lys Ser Ile Val Asp Glu Ile Met Lys
100 105 110
Ser Gly Phe Asp Gly Ile Phe Leu Ile Ala Thr Asn Pro Val Asp Val
115 120 125
Leu Thr Tyr Ala Val Gln Lys Phe Ser Gly Leu Pro Glu Asn Gln Val
130 135 140
Ile Gly Ser Gly Thr Ile Leu Asp Thr Ala Arg Phe Arg His Leu Leu
145 150 155 160
Ser Gln Glu Phe Asn Val Ser Pro Asn Ser Val His Gly Tyr Ile Ile
165 170 175
Gly Glu His Gly Asp Ser Glu Leu Ala Val Trp Ser Gly Thr Asn Ile
180 185 190
Ala Gly Asn Ser Leu Tyr Asp Ile Leu Asn Glu Asn Pro Glu Lys Gln
195 200 205
Lys Leu Ile Glu Glu Ile Phe Val Asn Thr Arg Asp Ala Ala Tyr Glu
210 215 220
Ile Ile Lys Ala Lys Gly Ala Thr Tyr Tyr Gly Val Ala Met Gly Leu
225 230 235 240
Met Arg Ile Ser Lys Ala Ile Leu Asn Asn Glu Asn Val Val Leu Thr
245 250 255
Val Ser Ala Lys Leu Asn Gly Glu Tyr Gly His Asp Asp Val Tyr Ile
260 265 270
Gly Val Pro Ala Ile Ile Asn Arg Asn Gly Ile Arg Glu Val Leu Glu
275 280 285
Thr Pro Leu Asn Thr Glu Glu Lys Glu Lys Phe Ala Lys Ser Val Glu
290 295 300
Thr Leu Lys Ala Ile Gln Thr Pro Phe Phe Ser
305 310 315
<210> 57
<211> 314
<212> PRT/БЕЛОК
<213> Bacillus thuringiensis
<400> 57
Met Lys Lys Gly Ile Asn Arg Val Val Leu Val Gly Thr Gly Ala Val
1 5 10 15
Gly Cys Ser Tyr Ala Tyr Ser Met Ile Asn Gln Gly Val Ala Glu Glu
20 25 30
Phe Val Leu Val Asp Val Asn Glu Ala Lys Ala Glu Gly Glu Ala Met
35 40 45
Asp Leu Ser His Ala Val Pro Phe Ser Pro Ser Pro Thr Lys Val Trp
50 55 60
Ser Gly Ser Tyr Ala Asp Cys Lys Asp Ala Asp Leu Val Val Ile Thr
65 70 75 80
Ala Gly Leu Pro Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val Glu
85 90 95
Lys Asn Thr Lys Ile Phe Lys Gln Ile Val Arg Gly Ile Met Asp Ser
100 105 110
Gly Phe Asp Gly Ile Phe Leu Ile Ala Thr Asn Pro Val Asp Ile Leu
115 120 125
Thr Tyr Val Thr Trp Lys Glu Ser Gly Leu Pro Lys Glu Arg Val Ile
130 135 140
Gly Ser Gly Thr Thr Leu Asp Ser Ala Arg Phe Arg Tyr Met Leu Gly
145 150 155 160
Asp Tyr Leu Asp Val Asp Pro Arg Asn Val His Ala Tyr Ile Val Gly
165 170 175
Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser His Ala Thr Ile Gly
180 185 190
Val Gln Lys Leu Glu Thr Ile Leu Ala Asn Asn Glu Gln Tyr Lys Gln
195 200 205
Glu Asp Leu Asp Lys Ile Phe Glu Asn Val Arg Asp Ala Ala Tyr His
210 215 220
Ile Ile Glu Arg Lys Gly Ala Thr Tyr Tyr Gly Ile Gly Met Ser Leu
225 230 235 240
Leu Arg Val Thr Lys Ala Ile Leu Asn Asn Glu Asn Ser Val Leu Thr
245 250 255
Val Ser Ala Tyr Leu Glu Gly Gln Tyr Gly Glu Lys Asp Ala Tyr Val
260 265 270
Gly Val Pro Ala Val Ile Asn Arg Glu Gly Val Arg Glu Ile Val Glu
275 280 285
Leu Glu Leu Asn Glu Glu Glu Lys Ala Lys Phe Ala His Ser Val Lys
290 295 300
Val Leu Lys Glu Thr Met Ala Pro Val Leu
305 310
<210> 58
<211> 314
<212> PRT/БЕЛОК
<213> Bacillus thuringiensis
<400> 58
Met Lys Lys Gly Ile Asn Arg Val Val Leu Val Gly Thr Gly Ala Val
1 5 10 15
Gly Cys Ser Tyr Ala Tyr Ser Met Ile Asn Gln Gly Val Ala Glu Glu
20 25 30
Phe Val Leu Val Asp Val Asn Glu Ala Lys Ala Glu Gly Glu Ala Met
35 40 45
Asp Leu Ser His Ala Val Pro Phe Ser Pro Ser Pro Thr Lys Val Trp
50 55 60
Ser Gly Ser Tyr Ala Asp Cys Lys Asp Ala Asp Leu Val Val Ile Thr
65 70 75 80
Ala Gly Leu Pro Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val Glu
85 90 95
Lys Asn Thr Lys Ile Phe Lys Gln Ile Val Arg Gly Ile Met Asp Ser
100 105 110
Gly Phe Asp Gly Ile Phe Leu Ile Ala Thr Asn Pro Val Asp Ile Leu
115 120 125
Thr Tyr Val Thr Trp Lys Glu Ser Gly Leu Pro Lys Glu Arg Val Ile
130 135 140
Gly Ser Gly Thr Thr Leu Asp Ser Ala Arg Phe Arg Tyr Met Leu Gly
145 150 155 160
Asp Tyr Leu Asp Val Asp Pro Arg Asn Val His Ala Tyr Ile Val Gly
165 170 175
Glu His Gly Asp Thr Glu Leu Pro Val Trp Ser His Ala Thr Ile Gly
180 185 190
Val Gln Lys Leu Glu Thr Ile Leu Ala Asn Asn Glu Gln Tyr Asn Gln
195 200 205
Glu Asp Leu Asp Lys Ile Phe Glu Asn Val Arg Asp Ala Ala Tyr His
210 215 220
Ile Ile Glu Arg Lys Gly Ala Thr Tyr Tyr Gly Ile Gly Met Ser Leu
225 230 235 240
Leu Arg Val Thr Lys Ala Ile Leu Asn Asn Glu Asn Ser Val Leu Thr
245 250 255
Val Ser Ala Tyr Leu Glu Gly Gln Tyr Gly Glu Lys Asp Ala Tyr Val
260 265 270
Gly Val Pro Ala Val Ile Asn Arg Glu Gly Val Arg Glu Ile Val Glu
275 280 285
Leu Glu Leu Asn Glu Asp Glu Lys Ala Lys Phe Ala His Ser Val Lys
290 295 300
Val Leu Lys Glu Thr Met Ala Pro Val Leu
305 310
<210> 59
<211> 320
<212> PRT/БЕЛОК
<213> Bacillus mycoides
<400> 59
Met Lys Gly Glu Ile Asn Met Lys Lys Gly Ile Asn Arg Val Val Leu
1 5 10 15
Val Gly Thr Gly Ala Val Gly Cys Ser Tyr Ala Tyr Ser Met Ile Asn
20 25 30
Gln Gly Val Ala Glu Glu Phe Val Leu Val Asp Val Asn Glu Ala Lys
35 40 45
Ala Glu Gly Glu Ala Met Asp Leu Ser His Ala Val Pro Phe Ser Pro
50 55 60
Ala Pro Thr Lys Val Trp Ser Gly Ser Tyr Ala Asp Cys Lys Asp Ala
65 70 75 80
Asp Leu Val Val Ile Thr Ala Gly Leu Pro Gln Lys Pro Gly Glu Thr
85 90 95
Arg Leu Asp Leu Val Glu Lys Asn Thr Lys Ile Phe Lys Gln Ile Val
100 105 110
Arg Gly Ile Met Asp Ser Gly Phe Asp Gly Ile Phe Leu Ile Ala Thr
115 120 125
Asn Pro Val Asp Ile Leu Thr Tyr Val Thr Trp Lys Glu Ser Gly Leu
130 135 140
Pro Lys Glu Arg Val Ile Gly Ser Gly Thr Thr Leu Asp Ser Ala Arg
145 150 155 160
Phe Arg Tyr Met Leu Gly Asp Tyr Leu Asp Val Asp Pro Arg Asn Val
165 170 175
His Ala Tyr Ile Val Gly Glu His Gly Asp Thr Glu Leu Pro Val Trp
180 185 190
Ser His Ala Thr Val Gly Val Gln Lys Leu Glu Thr Ile Leu Ala Asn
195 200 205
Asn Glu Gln Tyr Asn Gln Glu Asp Leu Asp Lys Ile Phe Glu Asn Val
210 215 220
Arg Asp Ala Ala Tyr His Ile Ile Glu Arg Lys Gly Ala Thr Tyr Tyr
225 230 235 240
Gly Ile Gly Met Ser Leu Leu Arg Val Thr Lys Ala Ile Leu Ser Asn
245 250 255
Glu Asn Ser Val Leu Thr Val Ser Ala Tyr Leu Glu Gly Gln Tyr Gly
260 265 270
Glu Lys Asp Ala Phe Val Gly Val Pro Ala Val Ile Asn Arg Glu Gly
275 280 285
Val Arg Glu Ile Val Glu Leu Glu Leu Asn Glu Glu Glu Lys Ala Lys
290 295 300
Phe Ala His Ser Val Lys Val Leu Lys Glu Thr Met Ala Pro Val Leu
305 310 315 320
<210> 60
<211> 312
<212> PRT/БЕЛОК
<213> Lactobacillus rhamnosus
<400> 60
Met Gln His Ser Gly Asn Ile Ile Leu Ile Gly Asp Gly Ala Ile Gly
1 5 10 15
Ser Ser Phe Ala Phe Asn Cys Leu Thr Thr Gly Val Gly Gln Ser Leu
20 25 30
Gly Ile Ile Asp Val Asn Glu Lys Arg Val Gln Gly Asp Val Glu Asp
35 40 45
Leu Ser Asp Ala Leu Pro Tyr Thr Ser Gln Lys Asn Ile Tyr Ala Ala
50 55 60
Ser Tyr Glu Asp Cys Lys Tyr Ala Asp Ile Ile Val Ile Thr Ala Gly
65 70 75 80
Ile Ala Gln Lys Pro Gly Gln Thr Arg Leu Glu Leu Leu Ser Ile Asn
85 90 95
Ala Lys Ile Ile Lys Glu Ile Thr His Asn Ile Met Ala Ser Gly Phe
100 105 110
Asn Gly Phe Ile Leu Val Ala Ser Asn Pro Val Asp Val Leu Ala Glu
115 120 125
Leu Val Leu Glu Glu Ser Gly Leu Pro Arg Asn Gln Val Leu Gly Ser
130 135 140
Gly Thr Ala Leu Asp Ser Ala Arg Leu Arg Ser Glu Ile Gly Leu Arg
145 150 155 160
Tyr Asn Val Asp Ala Arg Ile Val His Gly Tyr Ile Met Gly Glu His
165 170 175
Gly Asp Ser Glu Phe Pro Val Trp Asp Tyr Thr Asn Ile Gly Gly Lys
180 185 190
Pro Ile Leu Asp Trp Ile Pro Lys Asn Arg Gln Ala Ser Asp Leu Ala
195 200 205
Glu Ile Ser His Arg Val Lys Thr Ala Ala Tyr Gly Ile Ile Glu Lys
210 215 220
Lys Gly Ala Thr Phe Tyr Gly Ile Ala Ala Ser Leu Thr Arg Leu Thr
225 230 235 240
Ser Ala Phe Leu Asn Asp Asp Arg Ala Ala Phe Ala Met Ser Val His
245 250 255
Leu Asp Gly Glu Tyr Gly Leu Ser Gly Val Ser Ile Gly Val Pro Val
260 265 270
Ile Leu Gly Ala Asn Gly Leu Glu Arg Ile Ile Glu Leu Asp Leu Asn
275 280 285
Ala Glu Asp His Lys Arg Leu Ala Asp Ser Ala Ala Ile Leu Lys Asp
290 295 300
Asn Leu Lys Lys Ala Gln Glu Ala
305 310
<210> 61
<211> 313
<212> PRT/БЕЛОК
<213> Lactobacillus fermentum
<400> 61
Met Ala Arg Lys Val Ala Val Val Gly Met Gly Asn Val Gly Ala Thr
1 5 10 15
Val Ala His Tyr Leu Val Ala Gly Gly Phe Thr Asp Asp Leu Val Leu
20 25 30
Ile Asp Pro Arg Glu Glu Lys Val Val Ala Asp Ala Val Asp Phe Glu
35 40 45
Asp Ala Met Ala Asn Leu Glu Tyr His Thr Asn Ile Phe Val Asn Asp
50 55 60
Tyr Glu Ala Leu Ala Asp Ala Asp Val Val Val Ser Ala Leu Gly Asn
65 70 75 80
Ile Lys Leu Gln Asp Asn Pro Asp Asp Asp Arg Phe Ala Glu Leu Pro
85 90 95
Tyr Thr Arg Val Gln Val Lys Lys Val Ala Thr Lys Leu Lys Glu Val
100 105 110
Gly Phe Asn Gly Ile Ile Val Ala Ile Thr Asn Pro Val Asp Val Ile
115 120 125
Thr Ser Leu Tyr Gln Glu Ile Thr Gly Leu Pro Lys Asn His Val Ile
130 135 140
Gly Thr Gly Thr Leu Leu Asp Ser Ala Arg Met Lys Arg Ala Val Ala
145 150 155 160
Lys Lys Leu Asn Leu Asp Pro Arg Ser Val Ala Gly Tyr Asn Leu Gly
165 170 175
Glu His Gly Asn Ser Gln Phe Thr Ala Trp Ser Thr Val Arg Val Leu
180 185 190
Gly Lys Pro Ile Glu Gln Ile Ala Asp Gln Lys Gly Leu Asp Leu Val
195 200 205
Asp Leu Asp Lys Ala Ala Arg Glu Gly Gly Phe Ile Val Phe Arg Gly
210 215 220
Lys Lys Tyr Thr Ser Tyr Gly Val Ala Thr Ala Ala Val Arg Leu Val
225 230 235 240
Asn Thr Ile Leu Ser Asn Ala Leu Thr Glu Leu Pro Val Ser Asn Tyr
245 250 255
Arg Glu Glu Tyr Gly Val Tyr Leu Ser Tyr Pro Ala Val Val Gly Arg
260 265 270
Asp Gly Ile Val Glu Gln Cys Gln Leu Asp Leu Thr Ala Glu Glu Leu
275 280 285
Gln Lys Leu Gln Val Ser Ala Asp Phe Ile Lys Gln Lys Phe Ala Glu
290 295 300
Ser Leu Glu Ala Ala Asp Gln Glu Asp
305 310
<210> 62
<211> 307
<212> PRT/БЕЛОК
<213> Lactobacillus delbrueckii
<400> 62
Met Ser Arg Lys Val Leu Leu Val Gly Asp Gly Ala Val Gly Ser Asn
1 5 10 15
Phe Ala Asn Asp Leu Leu Gln Thr Thr Arg Val Asp Glu Leu Val Ile
20 25 30
Cys Asp Leu Asn Lys Asp Arg Ala Ala Gly Asp Cys Leu Asp Leu Glu
35 40 45
Asp Met Thr Tyr Phe Thr Gly Gln Thr Lys Leu Arg Ala Gly Asp Tyr
50 55 60
Ser Asp Ala Ala Asp Ala Asp Val Val Val Ile Thr Ala Gly Val Pro
65 70 75 80
Arg Lys Pro Gly Glu Ser Arg Leu Asp Leu Ile Lys Lys Asn Glu Ala
85 90 95
Ile Leu Arg Ser Ile Val Asp Pro Val Val Ala Ser Gly Phe Ser Gly
100 105 110
Ile Phe Val Val Ser Ala Asn Pro Val Asp Ile Leu Thr Thr Leu Thr
115 120 125
Gln Lys Leu Ser Gly Phe Pro Lys Lys Arg Val Ile Gly Thr Gly Thr
130 135 140
Ser Leu Asp Ser Ala Ser Leu Arg Val Glu Leu Ala Lys Arg Leu Gln
145 150 155 160
Val Pro Ile Glu Ser Val Asn Ala Trp Val Leu Gly Glu His Gly Asp
165 170 175
Ser Ser Phe Glu Asn Phe Ser Ser Ala Val Val Asn Gly Lys Pro Leu
180 185 190
Leu Asp Tyr Pro Gly Met Thr Glu Ala Ala Leu Asp Glu Ile Glu Ala
195 200 205
His Val Arg Glu Lys Gly Ser Glu Ile Ile Val Lys Lys Gly Ala Thr
210 215 220
Tyr Tyr Gly Val Ala Met Met Leu Ala Lys Ile Val Thr Ala Ile Leu
225 230 235 240
Glu Asn Asn Asp Leu Ala Leu Pro Leu Ser Ala Pro Leu His Gly Glu
245 250 255
Tyr Gly Ile Lys Asp Glu Ile Tyr Leu Gly Thr Leu Ala Ile Ile Asn
260 265 270
Gly Gln Gly Ile Ser His Val Leu Glu Leu Pro Leu Asn Asp Ser Glu
275 280 285
Leu Ala Lys Met Arg Ala Ser Ala Ala Thr Ile Lys Ala Thr Leu Asp
290 295 300
Ser Leu Gly
305
<210> 63
<211> 325
<212> PRT/БЕЛОК
<213> Lactococcus lactis
<400> 63
Met Ala Asp Lys Gln Arg Lys Lys Val Ile Leu Val Gly Asp Gly Ala
1 5 10 15
Val Gly Ser Ser Tyr Ala Phe Ala Leu Val Asn Gln Gly Ile Ala Gln
20 25 30
Glu Leu Gly Ile Val Asp Leu Phe Lys Glu Lys Thr Gln Gly Asp Ala
35 40 45
Glu Asp Leu Ser His Ala Leu Ala Phe Thr Ser Pro Lys Lys Ile Tyr
50 55 60
Ser Ala Asp Tyr Ser Asp Ala Ser Asp Ala Asp Leu Val Val Leu Thr
65 70 75 80
Ser Gly Ala Pro Gln Lys Pro Gly Glu Thr Arg Leu Asp Leu Val Glu
85 90 95
Lys Asn Leu Arg Ile Thr Lys Asp Val Val Thr Lys Ile Val Ala Ser
100 105 110
Gly Phe Lys Gly Ile Phe Leu Val Ala Ala Asn Pro Val Asp Ile Leu
115 120 125
Thr Tyr Ala Thr Trp Lys Phe Ser Gly Phe Pro Lys Asn Arg Val Val
130 135 140
Gly Ser Gly Thr Ser Leu Asp Thr Ala Arg Phe Arg Gln Ala Leu Ala
145 150 155 160
Glu Lys Val Asp Val Asp Ala Arg Ser Ile His Ala Tyr Ile Met Gly
165 170 175
Glu His Gly Asp Ser Glu Phe Ala Val Trp Ser His Ala Asn Val Ala
180 185 190
Gly Val Lys Leu Glu Gln Trp Phe Gln Glu Asn Asp Tyr Leu Asn Glu
195 200 205
Ala Glu Ile Val Glu Leu Phe Glu Ser Val Arg Asp Ala Ala Tyr Ser
210 215 220
Ile Ile Ala Lys Lys Gly Ala Thr Phe Tyr Gly Val Ala Val Ala Leu
225 230 235 240
Ala Arg Ile Thr Lys Ala Ile Leu Asp Asp Glu His Ala Val Leu Pro
245 250 255
Val Ser Val Phe Gln Asp Gly Gln Tyr Gly Val Ser Asp Cys Tyr Leu
260 265 270
Gly Gln Pro Ala Val Val Gly Ala Glu Gly Val Val Asn Pro Ile His
275 280 285
Ile Pro Leu Asn Asp Ala Glu Met Gln Lys Met Glu Ala Ser Gly Ala
290 295 300
Gln Leu Lys Ala Ile Ile Asp Glu Ala Phe Ala Lys Glu Glu Phe Ala
305 310 315 320
Ser Ala Val Lys Asn
325
<210> 64
<211> 1014
<212> DNA/ДНК
<213> ЛДГ Lactobacillus helveticus
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(1014)
<400> 64
atg aca aag gtt ttt gct tac gct att cga aaa gac gaa gaa cca ttc 48
Met Thr Lys Val Phe Ala Tyr Ala Ile Arg Lys Asp Glu Glu Pro Phe
1 5 10 15
ttg aat gaa tgg aag gaa gct cac aag gat atc gat gtt gat tac act 96
Leu Asn Glu Trp Lys Glu Ala His Lys Asp Ile Asp Val Asp Tyr Thr
20 25 30
gat aaa ctt ttg act cct gaa act gct aag cta gct aag ggt gct gac 144
Asp Lys Leu Leu Thr Pro Glu Thr Ala Lys Leu Ala Lys Gly Ala Asp
35 40 45
ggt gtt gtt gtt tac caa caa tta gac tac act gca gat act ctt caa 192
Gly Val Val Val Tyr Gln Gln Leu Asp Tyr Thr Ala Asp Thr Leu Gln
50 55 60
gct tta gca gac gct ggc gta act aag atg tca tta cgt aac gtt ggt 240
Ala Leu Ala Asp Ala Gly Val Thr Lys Met Ser Leu Arg Asn Val Gly
65 70 75 80
gtt gac aac att gat atg gac aag gct aag gaa tta ggt ttc caa att 288
Val Asp Asn Ile Asp Met Asp Lys Ala Lys Glu Leu Gly Phe Gln Ile
85 90 95
acc aat gtt cct gtt tac tca cca aac gct att gct gaa cat gct gct 336
Thr Asn Val Pro Val Tyr Ser Pro Asn Ala Ile Ala Glu His Ala Ala
100 105 110
att cag gct gca cgt gta tta cgt caa gac aag cgc atg gac gaa aag 384
Ile Gln Ala Ala Arg Val Leu Arg Gln Asp Lys Arg Met Asp Glu Lys
115 120 125
atg gct aaa cgt gac tta cgt tgg gca cca act atc ggc cgt gaa gtt 432
Met Ala Lys Arg Asp Leu Arg Trp Ala Pro Thr Ile Gly Arg Glu Val
130 135 140
cgt gac caa gtt gtc ggt gtt gtt ggt act ggt cac att ggt caa gta 480
Arg Asp Gln Val Val Gly Val Val Gly Thr Gly His Ile Gly Gln Val
145 150 155 160
ttt atg cgt att atg gaa ggt ttc ggt gca aag gtt att gct tac gat 528
Phe Met Arg Ile Met Glu Gly Phe Gly Ala Lys Val Ile Ala Tyr Asp
165 170 175
atc ttc aag aac cca gaa ctt gaa aag aag ggt tac tac gtt gac tca 576
Ile Phe Lys Asn Pro Glu Leu Glu Lys Lys Gly Tyr Tyr Val Asp Ser
180 185 190
ctt gac gac ttg tac aag caa gct gat gta att tca ctt cac gta cca 624
Leu Asp Asp Leu Tyr Lys Gln Ala Asp Val Ile Ser Leu His Val Pro
195 200 205
gat gtt cca gct aac gtt cac atg atc aac gac aag tca atc gct gaa 672
Asp Val Pro Ala Asn Val His Met Ile Asn Asp Lys Ser Ile Ala Glu
210 215 220
atg aaa gac ggc gtt gta att gta aac tgc tca cgt ggt cga ctt gtt 720
Met Lys Asp Gly Val Val Ile Val Asn Cys Ser Arg Gly Arg Leu Val
225 230 235 240
gac act gac gct gta atc cgt ggt ttg gac tca ggc aag atc ttc ggc 768
Asp Thr Asp Ala Val Ile Arg Gly Leu Asp Ser Gly Lys Ile Phe Gly
245 250 255
ttc gtt atg gat act tac gaa gac gaa gtt ggt gta ttt aac aag gat 816
Phe Val Met Asp Thr Tyr Glu Asp Glu Val Gly Val Phe Asn Lys Asp
260 265 270
tgg gaa ggt aaa gaa ttc cca gac aag cgt ttg gca gac tta att gat 864
Trp Glu Gly Lys Glu Phe Pro Asp Lys Arg Leu Ala Asp Leu Ile Asp
275 280 285
cgt cca aac gta ttg gta act cca cac acc gcc ttc tac act act cac 912
Arg Pro Asn Val Leu Val Thr Pro His Thr Ala Phe Tyr Thr Thr His
290 295 300
gct gta cgt aac atg gtt gtt aag gca ttc aac aac aac ttg aag tta 960
Ala Val Arg Asn Met Val Val Lys Ala Phe Asn Asn Asn Leu Lys Leu
305 310 315 320
atc aac ggc gaa aag cca gat tct cca gtt gct ttg aac aag aac aag 1008
Ile Asn Gly Glu Lys Pro Asp Ser Pro Val Ala Leu Asn Lys Asn Lys
325 330 335
ttt taa 1014
Phe
<210> 65
<211> 337
<212> PRT/БЕЛОК
<213> ЛДГ Lactobacillus helveticus
<400> 65
Met Thr Lys Val Phe Ala Tyr Ala Ile Arg Lys Asp Glu Glu Pro Phe
1 5 10 15
Leu Asn Glu Trp Lys Glu Ala His Lys Asp Ile Asp Val Asp Tyr Thr
20 25 30
Asp Lys Leu Leu Thr Pro Glu Thr Ala Lys Leu Ala Lys Gly Ala Asp
35 40 45
Gly Val Val Val Tyr Gln Gln Leu Asp Tyr Thr Ala Asp Thr Leu Gln
50 55 60
Ala Leu Ala Asp Ala Gly Val Thr Lys Met Ser Leu Arg Asn Val Gly
65 70 75 80
Val Asp Asn Ile Asp Met Asp Lys Ala Lys Glu Leu Gly Phe Gln Ile
85 90 95
Thr Asn Val Pro Val Tyr Ser Pro Asn Ala Ile Ala Glu His Ala Ala
100 105 110
Ile Gln Ala Ala Arg Val Leu Arg Gln Asp Lys Arg Met Asp Glu Lys
115 120 125
Met Ala Lys Arg Asp Leu Arg Trp Ala Pro Thr Ile Gly Arg Glu Val
130 135 140
Arg Asp Gln Val Val Gly Val Val Gly Thr Gly His Ile Gly Gln Val
145 150 155 160
Phe Met Arg Ile Met Glu Gly Phe Gly Ala Lys Val Ile Ala Tyr Asp
165 170 175
Ile Phe Lys Asn Pro Glu Leu Glu Lys Lys Gly Tyr Tyr Val Asp Ser
180 185 190
Leu Asp Asp Leu Tyr Lys Gln Ala Asp Val Ile Ser Leu His Val Pro
195 200 205
Asp Val Pro Ala Asn Val His Met Ile Asn Asp Lys Ser Ile Ala Glu
210 215 220
Met Lys Asp Gly Val Val Ile Val Asn Cys Ser Arg Gly Arg Leu Val
225 230 235 240
Asp Thr Asp Ala Val Ile Arg Gly Leu Asp Ser Gly Lys Ile Phe Gly
245 250 255
Phe Val Met Asp Thr Tyr Glu Asp Glu Val Gly Val Phe Asn Lys Asp
260 265 270
Trp Glu Gly Lys Glu Phe Pro Asp Lys Arg Leu Ala Asp Leu Ile Asp
275 280 285
Arg Pro Asn Val Leu Val Thr Pro His Thr Ala Phe Tyr Thr Thr His
290 295 300
Ala Val Arg Asn Met Val Val Lys Ala Phe Asn Asn Asn Leu Lys Leu
305 310 315 320
Ile Asn Gly Glu Lys Pro Asp Ser Pro Val Ala Leu Asn Lys Asn Lys
325 330 335
Phe
<210> 66
<211> 996
<212> DNA/ДНК
<213> ЛДГ Leuconostoc mesenteroides
<220>
<221> CDS/Кодирующие последовательности
<222> (1)..(996)
<400> 66
atg aag att ttt gct tac ggc att cgt gat gat gaa aag cca tca ctt 48
Met Lys Ile Phe Ala Tyr Gly Ile Arg Asp Asp Glu Lys Pro Ser Leu
1 5 10 15
gaa gaa tgg aaa gcg gct aac cca gag att gaa gtg gac tac aca caa 96
Glu Glu Trp Lys Ala Ala Asn Pro Glu Ile Glu Val Asp Tyr Thr Gln
20 25 30
gag cta ttg aca cct gaa aca gtt aag ttg gct gag gga tca gat tca 144
Glu Leu Leu Thr Pro Glu Thr Val Lys Leu Ala Glu Gly Ser Asp Ser
35 40 45
gct gtt gtt tac caa caa ctg gac tat aca cgt gaa aca ttg aca gct 192
Ala Val Val Tyr Gln Gln Leu Asp Tyr Thr Arg Glu Thr Leu Thr Ala
50 55 60
tta gct aac gtt ggt ggt act aac ttg tca ttg cgt aac gtt ggt aca 240
Leu Ala Asn Val Gly Gly Thr Asn Leu Ser Leu Arg Asn Val Gly Thr
65 70 75 80
gat aac att gat ttt gat gca gca cgt gaa ttt aac ttt aac att tca 288
Asp Asn Ile Asp Phe Asp Ala Ala Arg Glu Phe Asn Phe Asn Ile Ser
85 90 95
aat gtt cct gtt tat tca cca aat gct att gca gaa cac tca atg att 336
Asn Val Pro Val Tyr Ser Pro Asn Ala Ile Ala Glu His Ser Met Ile
100 105 110
caa tta tct cgt ttg cta cgt cgc acg aaa gca ttg gat gcc aaa att 384
Gln Leu Ser Arg Leu Leu Arg Arg Thr Lys Ala Leu Asp Ala Lys Ile
115 120 125
gct aag cac gac ttg cgc tgg gca cca aca att gga cgt gaa atg cgt 432
Ala Lys His Asp Leu Arg Trp Ala Pro Thr Ile Gly Arg Glu Met Arg
130 135 140
atg caa aca gtt ggt gtt att ggt aca ggc cat att ggc cgt gtt gct 480
Met Gln Thr Val Gly Val Ile Gly Thr Gly His Ile Gly Arg Val Ala
145 150 155 160
att aac att ttg aaa ggc ttt ggg gca aag gtt att gct tat gat aag 528
Ile Asn Ile Leu Lys Gly Phe Gly Ala Lys Val Ile Ala Tyr Asp Lys
165 170 175
tac cca aat gct gaa ttg caa gca gaa ggt ttg tac gtt gac aca tta 576
Tyr Pro Asn Ala Glu Leu Gln Ala Glu Gly Leu Tyr Val Asp Thr Leu
180 185 190
gac gaa tta tat gca caa gct gat gca att tca ttg tat gtt cct ggt 624
Asp Glu Leu Tyr Ala Gln Ala Asp Ala Ile Ser Leu Tyr Val Pro Gly
195 200 205
gtg cct gaa aac cat cat cta atc aat gca gag gct att gct aag atg 672
Val Pro Glu Asn His His Leu Ile Asn Ala Glu Ala Ile Ala Lys Met
210 215 220
aat gat ggc gtg gtt atc atg aat gct gcg cgt ggt aat ttg atg gac 720
Asn Asp Gly Val Val Ile Met Asn Ala Ala Arg Gly Asn Leu Met Asp
225 230 235 240
att gat gct att att gat ggt ttg aat tct ggt aag att tca gac ttc 768
Ile Asp Ala Ile Ile Asp Gly Leu Asn Ser Gly Lys Ile Ser Asp Phe
245 250 255
ggt atg gac gtt tat gaa aat gaa gtt ggc ttg ttc aat gaa gat tgg 816
Gly Met Asp Val Tyr Glu Asn Glu Val Gly Leu Phe Asn Glu Asp Trp
260 265 270
tct ggt aaa gaa ttc cca gat gct aag att gct gac ttg att tca cgc 864
Ser Gly Lys Glu Phe Pro Asp Ala Lys Ile Ala Asp Leu Ile Ser Arg
275 280 285
gaa aat gta ttg gtt acg cca cat acg gct ttc tat aca act aaa gct 912
Glu Asn Val Leu Val Thr Pro His Thr Ala Phe Tyr Thr Thr Lys Ala
290 295 300
gtt cta gaa atg gtt cac caa tca ttt gat gca gca gtt gct ttc gcc 960
Val Leu Glu Met Val His Gln Ser Phe Asp Ala Ala Val Ala Phe Ala
305 310 315 320
aaa ggt gag aag cca gct att gct gtt gaa tat taa 996
Lys Gly Glu Lys Pro Ala Ile Ala Val Glu Tyr
325 330
<210> 67
<211> 331
<212> PRT/БЕЛОК
<213> ЛДГ Leuconostoc mesenteroides
<400> 67
Met Lys Ile Phe Ala Tyr Gly Ile Arg Asp Asp Glu Lys Pro Ser Leu
1 5 10 15
Glu Glu Trp Lys Ala Ala Asn Pro Glu Ile Glu Val Asp Tyr Thr Gln
20 25 30
Glu Leu Leu Thr Pro Glu Thr Val Lys Leu Ala Glu Gly Ser Asp Ser
35 40 45
Ala Val Val Tyr Gln Gln Leu Asp Tyr Thr Arg Glu Thr Leu Thr Ala
50 55 60
Leu Ala Asn Val Gly Gly Thr Asn Leu Ser Leu Arg Asn Val Gly Thr
65 70 75 80
Asp Asn Ile Asp Phe Asp Ala Ala Arg Glu Phe Asn Phe Asn Ile Ser
85 90 95
Asn Val Pro Val Tyr Ser Pro Asn Ala Ile Ala Glu His Ser Met Ile
100 105 110
Gln Leu Ser Arg Leu Leu Arg Arg Thr Lys Ala Leu Asp Ala Lys Ile
115 120 125
Ala Lys His Asp Leu Arg Trp Ala Pro Thr Ile Gly Arg Glu Met Arg
130 135 140
Met Gln Thr Val Gly Val Ile Gly Thr Gly His Ile Gly Arg Val Ala
145 150 155 160
Ile Asn Ile Leu Lys Gly Phe Gly Ala Lys Val Ile Ala Tyr Asp Lys
165 170 175
Tyr Pro Asn Ala Glu Leu Gln Ala Glu Gly Leu Tyr Val Asp Thr Leu
180 185 190
Asp Glu Leu Tyr Ala Gln Ala Asp Ala Ile Ser Leu Tyr Val Pro Gly
195 200 205
Val Pro Glu Asn His His Leu Ile Asn Ala Glu Ala Ile Ala Lys Met
210 215 220
Asn Asp Gly Val Val Ile Met Asn Ala Ala Arg Gly Asn Leu Met Asp
225 230 235 240
Ile Asp Ala Ile Ile Asp Gly Leu Asn Ser Gly Lys Ile Ser Asp Phe
245 250 255
Gly Met Asp Val Tyr Glu Asn Glu Val Gly Leu Phe Asn Glu Asp Trp
260 265 270
Ser Gly Lys Glu Phe Pro Asp Ala Lys Ile Ala Asp Leu Ile Ser Arg
275 280 285
Glu Asn Val Leu Val Thr Pro His Thr Ala Phe Tyr Thr Thr Lys Ala
290 295 300
Val Leu Glu Met Val His Gln Ser Phe Asp Ala Ala Val Ala Phe Ala
305 310 315 320
Lys Gly Glu Lys Pro Ala Ile Ala Val Glu Tyr
325 330
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ПОЛУЧЕНИЯ L-АМИНОКИСЛОТ С ПОМОЩЬЮ КОРИНЕБАКТЕРИЙ С ПРИМЕНЕНИЕМ СИСТЕМЫ РАСЩЕПЛЕНИЯ ГЛИЦИНА | 2015 |
|
RU2713298C2 |
| КОНСТРУКЦИИ НУКЛЕИНОВОЙ КИСЛОТЫ И ВЕКТОРЫ ДЛЯ ГЕНОТЕРАПИИ ДЛЯ ПРИМЕНЕНИЯ ДЛЯ ЛЕЧЕНИЯ БОЛЕЗНИ ВИЛЬСОНА И ДРУГИХ СОСТОЯНИЙ | 2015 |
|
RU2745567C2 |
| СПОСОБ УПРАВЛЕНИЯ РИТМОМ СЕРДЦА И СОКРАЩЕНИЕМ ОТДЕЛЬНЫХ КАРДИОМИОЦИТОВ ПРИ ПОМОЩИ ТЕРМОГЕНЕТИКИ | 2022 |
|
RU2802995C1 |
| Вакцина против герпеса | 2019 |
|
RU2731073C1 |
| СПОСОБ ПРОДУЦИРОВАНИЯ ПОЧЕЧНЫХ КЛЕТОК-ПРЕДШЕСТВЕННИКОВ | 2016 |
|
RU2730861C2 |
| БИОСЕНСОРНАЯ СИСТЕМА ДЛЯ БЫСТРОГО ОБНАРУЖЕНИЯ ОПРЕДЕЛЯЕМЫХ КОМПОНЕНТОВ | 2016 |
|
RU2717658C2 |
| ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ ДЛЯ ЛЕЧЕНИЯ И/ИЛИ ПРЕДОТВРАЩЕНИЯ ЗЛОКАЧЕСТВЕННОЙ ОПУХОЛИ | 2016 |
|
RU2714205C2 |
| ИММУНОИНДУЦИРУЮЩЕЕ СРЕДСТВО | 2016 |
|
RU2744843C2 |
| Антитела против белка р17 ВИЧ-1 субтипа А | 2019 |
|
RU2727673C1 |
| СПОСОБ ПОЛУЧЕНИЯ ХИМИЧЕСКИХ ПРОДУКТОВ ТОНКОГО СИНТЕЗА С ПОМОЩЬЮ CORYNEBACTERIUM, СЕКРЕТИРУЮЩЕЙ МОДИФИЦИРОВАННЫЕ α-1,6-ГЛЮКОЗИДАЗЫ | 2018 |
|
RU2763317C2 |
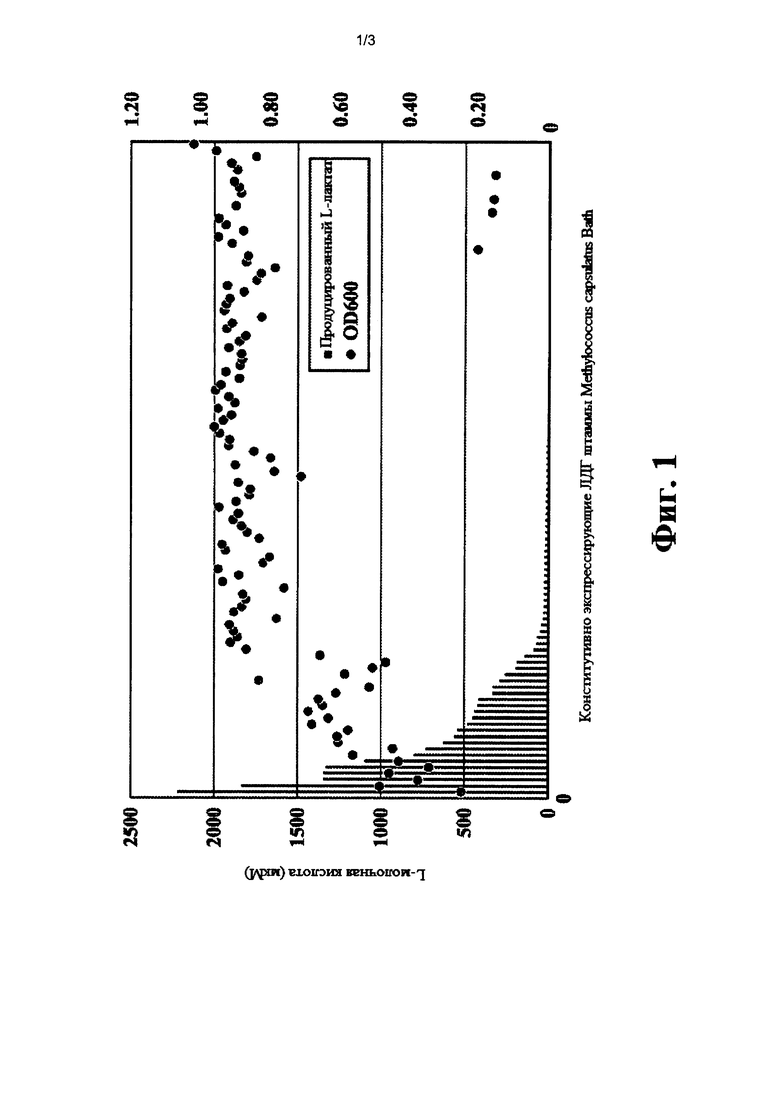
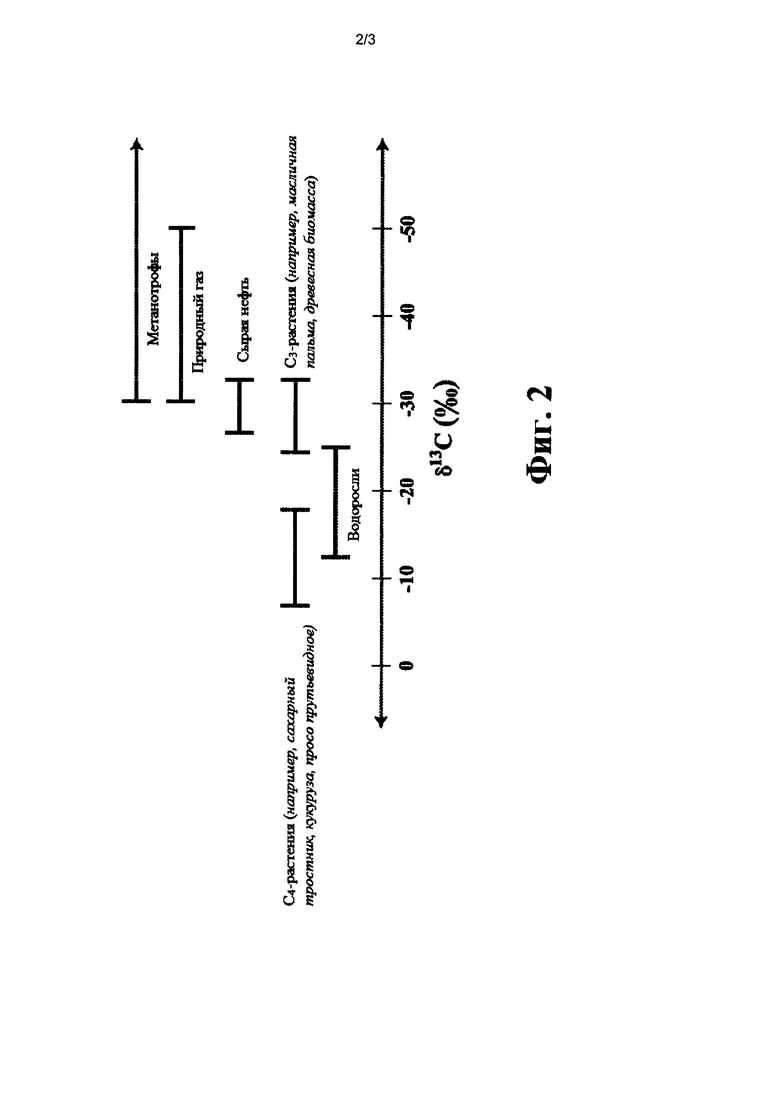
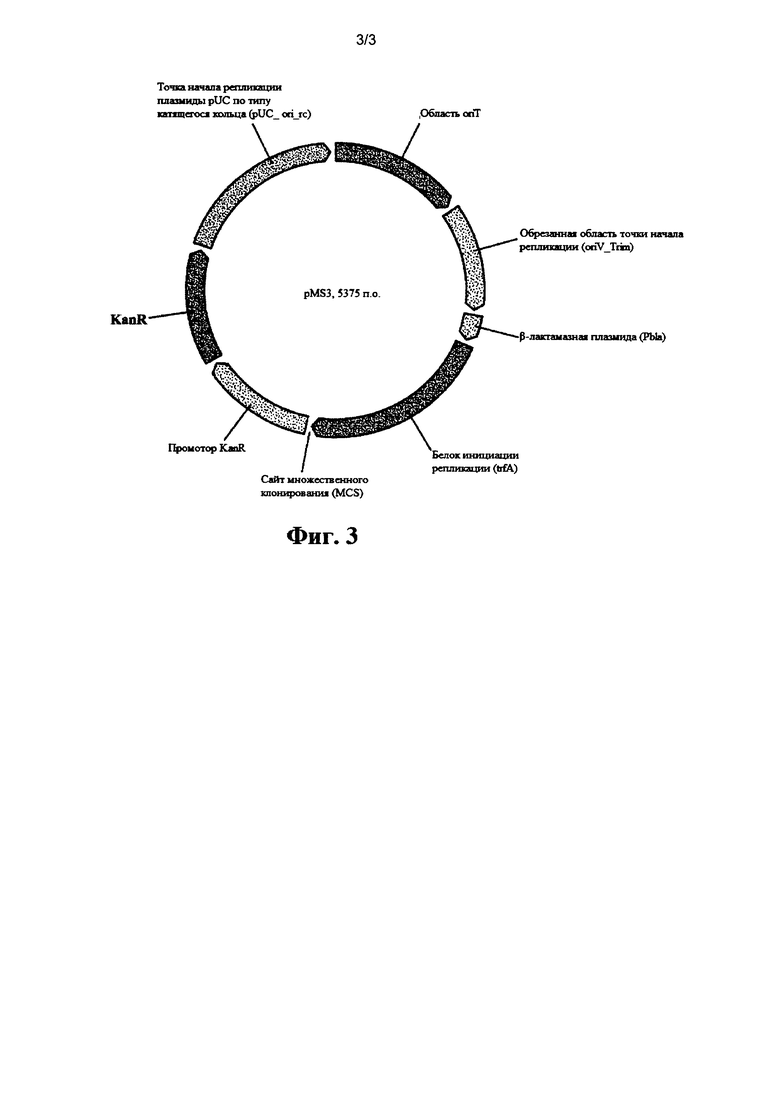
Изобретение относится к биотехнологии и представляет собой метанотрофную бактерию, содержащую гетерологичную нуклеиновую кислоту, кодирующую лактатдегидрогеназу (ЛДГ), причем указанная метанотрофная бактерия способна преобразовывать метан в лактат. Изобретение относится также к способу получения лактата, включающему культивирование указанной метанотрофной бактерии в присутствии углеродного сырья, содержащего метан, в условиях, достаточных для получения лактата, где указанная метанотрофная бактерия преобразует метан в лактат. Изобретение позволяет эффективно преобразовывать метан в лактат. 3 н. и 22 з.п. ф-лы, 3 ил., 3 табл., 2 пр.
1. Метанотрофная бактерия, содержащая гетерологичную нуклеиновую
кислоту, кодирующую лактатдегидрогеназу (ЛДГ), причем указанная метанотрофная бактерия способна преобразовывать метан в лактат.
2. Метанотрофная бактерия по п. 1, отличающаяся тем, что метан содержится в природном газе.
3. Метанотрофная бактерия по п. 1 или 2, отличающаяся тем, что указанная метанотрофная бактерия выбрана из группы, состоящей из метанотрофа I типа, метанотрофа II типа и метанотрофа X типа.
4. Метанотрофная бактерия по любому из пп. 1-3, отличающаяся тем, что указанная метанотрофная бактерия представляет собой факультативного метанотрофа или облигатного метанотрофа.
5. Метанотрофная бактерия по любому из пп. 1-4, отличающаяся тем, что указанная метанотрофная бактерия выбрана из группы, состоящей из Methylococcus, Methylomonas, Methylobacter, Methylosinus, Methylocystis, Methylomicrobium, Methanomonas, Methylocella, Methylacidiphilum, Methyloacida, Methylibium и Methylocapsa.
6. Метанотрофная бактерия по любому из пп. 1-4, отличающаяся тем, что указанная метанотрофная бактерия выбрана из группы, состоящей из Methylococcus capsulatus, шт. Bath, Methylomonas methanica 16a, Methylosinus trichosporium OB3b, Methylosinus sporium, Methylocystis parvus, Methylomonas methanica, Methylomonas albus, Methylobacter capsulatus, Methylobacterium organophilum, Methylomonas sp AJ-3670, Methylocella silvestris, Methylocella palustris, Methylocella tundrae, Methylocystis daltona, штамм SB2, Methylocystis bryophila, Methylocapsa aurea KYG, Methylacidiphilum infernorum, Methylacidiphilum fumariolicum, Methyloacida kamchatkensis, Methylibium petroleiphilum и Methylomicrobium alcaliphilum.
7. Метанотрофная бактерия по любому из пп. 1-6, отличающаяся тем, что указанная метанотрофная бактерия представляет собой Methylococcus capsulatus, шт. Bath или Methylosinus trichosporium ОВ3b.
8. Метанотрофная бактерия по любому из пп. 1-7, отличающаяся тем, что указанная гетерологичная нуклеиновая кислота кодирует лактатдегидрогеназу, содержащую аминокислотную последовательность, по меньшей мере на 85% идентичную эталонной аминокислотной последовательности, выбранной из группы, состоящей из последовательностей SEQ ID NO:34, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 36, 38, 40, 42, 44, 46, 48, 52, 53, 54, 55, 56, 57 и 58.
9. Метанотрофная бактерия по п. 8, отличающаяся тем, что указанная эталонная аминокислотная последовательность выбрана из группы, состоящей из последовательностей SEQ ID NO:34, 16 и 24.
10. Метанотрофная бактерия по любому из пп. 1-7, отличающаяся тем, что указанная гетерологичная нуклеиновая кислота кодирует лактатдегидрогеназу, имеющую аминокислотную последовательность, соответствующую аминокислотной последовательности, выбранной из группы, состоящей из последовательностей SEQ ID NO:34, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 36, 38, 40, 42, 44, 46, 48, 52, 53, 54, 55, 56, 57 и 58.
11. Метанотрофная бактерия по любому из пп. 1-7, отличающаяся тем, что указанная гетерологичная нуклеиновая кислота содержит последовательность нуклеиновой кислоты, выбранную из группы, состоящей из последовательностей SEQ ID NO:33, 1, 3, 5, 7, 9, 11, 13, 15, 17, 19, 21, 23, 25, 27, 29, 31, 35, 37, 39, 41, 43, 45 и 47.
12. Метанотрофная бактерия по любому из пп. 1-7, отличающаяся тем, что указанная молекула гетерологичной нуклеиновой кислоты кодирует ЛДГ из микроорганизма, выбранного из группы, состоящей из Listeria marthii, Actinomyces viscosus, Acinonyx jubatus, Archilochus colubris, Bacillus anthracis, Bacillus caldolyticus, Bacillus coagulans, Bacillus megaterium, Bacillus stearothermophilus (Q9p4b6) (также известного как Geobacillus stearothermophilus), Bacillus subtilis, Bacillus thuringiensis, Bacteroides pectinophilus, Bifidobacterium longum, Deinococcus radiodurans, Enterococcus faecalis, Enterococcus faecium, Kluyveromyces lactis, Kluyveromyces maxxianus, Lactobacillus acidophilus, Lactobacillus bulgaricus, Lactobacillus casei, Lactobacillus coryniformis sp. torquens, Lactobacillus delbrueckii (в том числе подвида bulgaricus), Lactobacillus fermentum, Lactobacillus helveticus, Lactobacillus johnsonii, Lactobacillus pentosus, Lactobacillus plantarum, Lactobacillus rhamnosus, Lactococcus lactis, Listeria monocytogenes, Plasmodium falciparum, Plasmodium ovale, Thermus thermophilus, Mus musculus, Oryctolagus cuniculus, Pediococcus acidilactici, Taeniopygia guttata, Rattus norvegicus, Rhizopus oryzae, Staphylococcus aureus, Streptococcus bovis, Streptococcus pasteurianus, Ruminococcus torques, Staphylococcus simiae, Staphylococcus vitulinus, Staphylococcus lentus, Macrococcus caseolyticus, Bacillus thuringiensis, серовариант konkukian, шт. 97-27, Bacillus thuringiensis, серологический вариант chinensis CT-43, и Bacillus mycoides.
13. Метанотрофная бактерия по любому из пп. 1-12, отличающаяся тем, что указанная последовательность гетерологичной нуклеиновой кислоты, кодирующая ЛДГ, кодон-оптимизирована для экспрессии в метанотрофной бактерии.
14. Метанотрофная бактерия по любому из пп. 1-13, отличающаяся тем, что указанная молекула гетерологичной нуклеиновой кислоты, кодирующая указанную лактатдегидрогеназу, функционально связана с индуцируемым промотором.
15. Метанотрофная бактерия по любому из пп. 1-14, отличающаяся тем, что указанная лактатдегидрогеназа представляет собой L-лактатдегидрогеназу.
16. Метанотрофная бактерия по любому из пп. 1-15, отличающаяся тем, что указанная лактатдегидрогеназа представляет собой D-лактатдегидрогеназу.
17. Способ получения лактата, включающий культивирование метанотрофной бактерии по любому из пп. 1-16 в присутствии углеродного сырья, содержащего метан, в условиях, достаточных для получения лактата, где указанная метанотрофная бактерия преобразует метан в лактат.
18. Способ по п. 17, отличающийся тем, что углеродное сырье представляет собой природный газ.
19. Способ по п. 17 или 18, отличающийся тем, что условия, достаточные для получения лактата, включают культивирование метанотрофной бактерии в ферментационной среде, которая содержит буфер.
20. Способ по любому из пп. 17-19, дополнительно включающий этап выделения лактата из метанотрофной бактерии.
21. Способ по любому из пп. 17-20, дополнительно включающий этап очистки лактата.
22. Способ по любому из пп. 17-21, отличающийся тем, что указанный лактат представляет собой L-лактат.
23. Лактатное соединение, содержащее одну или большее количество CH3C(OH)C(O)-групп, полученное способом по любому из пп. 17-22, причем δ13C указанной(ых) CH3C(OH)C(O)-групп(ы) составляет менее чем приблизительно -30‰.
24. Лактатное соединение по п. 23, отличающееся тем, что δ13C
CH3C(OH)C(O)-групп(ы) варьирует от приблизительно -70‰ до приблизительно -30‰.
25. Лактатное соединение по п. 24, отличающееся тем, что δ13C CH3C(OH)C(O)-групп(ы) варьирует от приблизительно -60‰ до приблизительно -40‰.
| ANGERMAYR SA et al.Engineering a cyanobacterial cell factory for production of lactic acid, Appl Environ Microbiol | |||
| Изложница с суживающимся книзу сечением и с вертикально перемещающимся днищем | 1924 |
|
SU2012A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Изложница с суживающимся книзу сечением и с вертикально перемещающимся днищем | 1924 |
|
SU2012A1 |
| EP1932910 A1, 18.06.2008 | |||
| RU 2003108880, 20.10.2004. | |||
Авторы
Даты
2020-01-10—Публикация
2014-06-18—Подача