ПЕРЕКРЕСТНАЯ ССЫЛКА
[0001] Настоящей заявке испрашивается приоритет по предварительной заявке США №62/783914, поданной 21 декабря 2018 года; предварительной заявке США №62/826827, поданной 29 марта 2019 года; предварительной заявке США №62/855379, поданной 31 мая 2019 года; и предварительной заявке США №62/891101, поданной 23 августа 2019 года, каждая из которых включена в настоящее описание в качестве ссылки в полном объеме.
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
[0001.1] Настоящая заявка содержит список последовательностей, который предоставлен в электронной форме в формате ASCII и включен в настоящее описание в качестве ссылки в полном объеме. Указанная копия ASCII, созданная 31 января 2020 года, названа 50401-735_601_SL.txt имеет размер 27306 байт.
УРОВЕНЬ ТЕХНИКИ
[0002] Главный комплекс гистосовместимости (MHC) представляет собой комплекс генов, кодирующий лейкоцитарные антигены человека (HLA). Гены HLA экспрессируются в качестве белковых гетеродимеров, которые экспонируются на поверхности клеток человека циркулирующим T-клеткам. Гены HLA являются в высокой степени полиморфными, что позволяет им тонко регулировать адаптивную иммунную систему. Адаптивные иммунные ответы основаны, частично, на способности T-клеток идентифицировать и устранять клетки, которые экспонируют ассоциированные с заболеванием пептидные антигены, связанные с гетеродимерами лейкоцитарого антигена человека (HLA).
[0003] У человека эндогенные и экзогенные белки могут процессироваться в пептиды протеасомой и цитозольными и эндосомальными/лизосомальными протеазами и пептидазами, и они презентируются двумя классами белков клеточной поверхности, кодируемых генами MHC. Эти белки клеточной поверхности называют лейкоцитарными антигенами человека (HLA класса I и класса II), и группу пептидов, которые связывают их и индуцируют иммунный ответ, называют эпитопами HLA. Эпитопы HLA являются ключевым компонентом, который позволяет иммунной системе выявлять сигналы опасности, такие как инфицирование патогенами и трансформация своего. CD4+ T-клетки распознают эпитопы MHC класса II (HLA-DR, HLA-DQ и HLA-DP), экспонированные на антигенпредставляющих клетках (APC), таких как дендритные клетки и макрофаги. Эндогенный процессинг и презентация лигандов HLA класса II является комплексным процессом и вовлекает различные шапероны и подгруппу ферментов, которые не являются хорошо охарактеризованными. Презентация пептидов HLA класса II активирует хелперные T-клетки, затем стимулируя дифференцировку B-клеток и продуцирование антител, а также ответы CTL. Активированные хелперные T-клетки также секретируют цитокины и хемокины, которые активируют и индуцируют дифференцировку других T-клеток.
[0004] Понимание предпочтений в отношении связывания пептидов у каждого гетеродимера HLA класса II является ключом к успешному прогнозированию того, какие антигены злокачественной опухоли или опухолеспецифические антигены вероятно будут индуцировать специфические для злокачественной опухоли или опухолеспецифические T-клеточные ответы. Существует потребность в идентификации и выделении определенных ассоциированных с HLA класса II пептидов (например, неоантигенных пептидов). Такая методология и выделенные молекулы являются пригодными, например, для разработки терапевтических средств, включая, но не ограничиваясь ими, терапевтические средства на иммунной основе.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
[0005] Способы и композиции, описанные в настоящем описании, применимы в широком диапазоне применений. Например, способы и композиции, описанные в настоящем описании, могут использоваться для идентификации иммуногенных антигенных пептидов и могут использоваться для разработки лекарственных средств, таких как лекарственные средства персонализированной медицины, и выделения и охарактеризации антигенспецифических T-клеток.
[0006] Ответы CD4+ T-клеток могут иметь противоопухолевую активность. Высокий уровень ответов CD4+ T-клеток может быть показан без использования прогнозирования для класса II (например, 60% эпитопов SLP в исследовании NeoVax (49% в NT-001, см. Ott et al., Nature, 2017 Jul 13;547(7662):217-221), и 48% мРНК-эпитопов в исследовании Biontech, см. Sahin et al., Nature, 2017 Jul 13;547(7662):222-226). Может не быть очевидно, презентируются ли эти эпитопы в нативных условиях (опухолью или фагоцитарными DC). Может быть желательным перевод высокого уровня CD4+ T-ответа в терапевтическую эффективность путем усовершенствования идентификации истинно презентируемых эпитопов, связываемых HLA класса II.
[0007] Роли генной экспрессии, ферментативного расщепления и предпочтения каскадов/локализации не могут быть достоверно количественно определены. Может быть неясно, является ли аутофагия (презентация HLA классу II опухолевыми клетками) или фагоцитоз (презентация HLA класса II опухолевых эпитопов посредством APC) более важным путем, хотя большинство существующих данных MS свидетельствует в пользу аутофагии. NetMHCIIpan может быть современным стандартом прогнозирования, однако его нельзя считать точным. Среди трех локусов HLA класса II (DR, DP и DQ), данные могут существовать только для определенных распространенных аллелей HLA-DR.
[0008] Для изучения правил презентации посредством HLA класса II могут использоваться различные подходы, включая стандартный в данной области и предлагаемый подход. Стандартный в данной области подход может включать измерение аффинности, которая может быть основой для средства прогнозирования NetMHCIIpan, обеспечивающего низкую производительность и требующего радиоактивных реагентов, и оно не учитывает роль процессинга. Предлагаемый подход может включать масс-спектрометрию, где данные для клеточных линий/тканей/опухолей могут помочь определить правила процессинга для аутофагии, и моноаллельная MS может позволить определение правил аллель-специфического связывания (данные мультиаллельной MS предположительно чрезмерно сложны для эффективного изучения (Bassani-Sternberg. MCP. 2018)).
[0009] Могут существовать различные способы подтверждения новых прогностических факторов для HLA класса II: подтверждение удержанных данных MS, которое может осуществляться по умолчанию; ретроспективные испытания вакцин (например, NT-001), где данные иммунного мониторинга могут оценивать пептидную нагрузку в вакцине на APC, а не презентацию опухоли, и данные могут быть понемногу распределены среди многих различных аллелей; биохимическое измерение аффинности, которое может быть организовано для получения измерений для дискордантно спрогнозированных пептидов (только для 2-3 аллелей); индукцию T-клеток, которая может быть организована для тестирования уровней, на которых предпочтительные согласно Neon и предпочтительные согласно NetMHCIIpan эпитопы индуцируют T-клеточные ответы ex vivo.
[0010] Для валидации посредством индукции T-клеток подход по умолчанию может включать оценку neoORF из TCGA, которые спрогнозированы дискордантно, где материалы для индукции могут включать APC и T-клетки здоровых доноров, и индукция и считывание могут осуществляться посредством SLP (~15-мерные пептиды). Случайные пептиды могут обеспечивать высокий уровень ответов и SLP могут быть в недостаточной степени учитывать процессинг. Возможные решения могут включать индукцию посредством мРНК.
[0011] Способы, описанные в настоящем описании, могут включать получение моноаллельных данных LC-MS/MS для обучения аллель-специфических машинных способов обучения для прогнозирования эпитопов. Такие способы могут включать повышение качества данных LC-MS/MS с использованием набора качественных показателей для точного удаления ложноположительных результатов, которые повышают эффективность модели прогнозирования; идентификацию аллель-специфических центров связывания HLA класса II из наборов данных LC-MS/MS для лигандома HLA; использование машинных алгоритмов обучения для улучшения прогнозирования лигандов и эпитопов HLA класса II; и/или идентификацию биологических переменных, которые влияют на презентацию лигандов HLA класса II и улучшают прогнозирование эпитопов HLA класса II, таких как экспрессия генов, способность к расщеплению, генное смещение, клеточная локализация и вторичная структура.
[0012] В рамках настоящего изобретения предусматривается способ, включающий: (a) обработку информации об аминокислотах для множества пептидных последовательностей-кандидатов с использованием машинно-обучаемой модели для прогнозирования презентации HLA для получения множества прогнозов презентации, где каждая пептидная последовательность-кандидат из множества пептидных последовательностей-кандидатов кодируется геномом или экзомом индивидуума, где множество прогнозов презентации включает прогноз презентации HLA для каждой из множества пептидных последовательностей-кандидатов, где каждый прогноз презентации HLA указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II в клетке индивидуума, может презентировать данную пептидную последовательность-кандидат из множества пептидных последовательностей-кандидатов, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательностях обучающих пептидов, идентифицированных посредством масс-спектрометрии в качестве презентируемых белком HLA, экспрессируемым в клетках для обучения; и (b) идентификацию на основе по меньшей мере множества прогнозов презентации пептидной последовательности из множества пептидных последовательностей в качестве презентируемых по меньшей мере одним из одного или более белков, кодируемых аллелем HLA класса II клетки индивидуума; где машинно-обучаемая модель для прогнозирования презентации пептидов HLA имеет положительную прогностическую величину (PPV) по меньшей мере 0,07 в соответствии со способом определения PPV презентации.
[0013] В рамках настоящего изобретения предусматривается способ, включающий: (a) обработку информации об аминокислотах для множества пептидных последовательностей, кодируемых геномом или экзомом индивидуума, с использованием машинно-обучаемой модели для прогнозирования связывания пептидов HLA с получением множества прогнозов связывания, где множество прогнозов связывания включает прогноз связывания HLA для каждой из множества пептидных последовательностей-кандидатов, причем каждый прогноз связывания указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II в клетке индивидуума связывается с данной пептидной последовательностью-кандидатом из множества пептидных последовательностей-кандидатов, где машинно-обучаемая модель для прогнозирования связывания пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательности пептидов, идентифицированных в качестве связывающихся с белком HLA класса II или аналогом HLA класса II; и (b) идентификацию на основе по меньшей мере множества прогнозов связывания пептидной последовательности из множества пептидных последовательностей, которая имеет вероятность, превышающую пороговую величину вероятности прогноза связывания, связываться по меньшей мере с одним из одного или более белков, кодируемых аллелем HLA класса II в клетке индивидуума; где машинно-обучаемая модель для прогнозирования связывания пептидов HLA имеет положительную прогностическую величину (PPV) по меньшей мере 0,1 в соответствии со способом определения PPV связывания.
[0014] В некоторых вариантах осуществления машинно-обучаемая модель для прогнозирования презентации пептидов посредством HLA обучена с использованием обучающих данных, включающих информацию о последовательности обучающих пептидов, идентифицированных посредством масс-спектрометрии в качестве презентируемых белком HLA, экспрессируемым в клетках для обучения.
[0015] В некоторых вариантах осуществления способ включает ранжирование, на основе прогнозов презентации, по меньшей мере двух пептидов, идентифицированных в качестве презентируемых по меньшей мере одним из одного или более белков, кодируемых аллелем HLA класса II, в клетке индивидуума.
[0016] В некоторых вариантах осуществления способ включает выбор одного или более пептидов из двух или более ранжированных пептидов.
[0017] В некоторых вариантах осуществления способ включает выбор одного или более пептидов из множества, которые были идентифицированы в качестве презентирующих по меньшей мере одним из одного или более белков, кодируемых аллелем HLA класса II, в клетке индивидуума.
[0018] В некоторых вариантах осуществления способ включает выбор одного или более пептидов из двух или более пептидов, ранжированных на основе прогнозов презентации.
[0019] В некоторых вариантах осуществления машинно-обучаемая модель для прогнозирования презентации пептидов HLA имеет положительную прогностическую величину (PPV) по меньшей мере 0,07, когда информация об аминокислотах для множества тестовых пептидных последовательностей обрабатывается для получения множества тестовых прогнозов презентации, причем каждый тестовый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II, в клетке индивидуума может презентировать данную тестовую пептидную последовательность из множества тестовых пептидных последовательностей, где множество тестовых пептидных последовательностей содержит по меньшей мере 500 тестовых пептидных последовательностей, содержащих (i) по меньшей мере одну из наиболее подходящих пептидных последовательностей, идентифицированных посредством масс-спектрометрии в качестве презентируемых белком HLA, экспрессируемым в клетках, и (ii) по меньшей мере 499 пептидных последовательностей-ловушек, содержащихся в белке, кодируемом геномом организма, где организм и индивидуум принадлежат одному виду, где множество тестовых последовательностей включает соотношение 1:499 для по меньшей мере одной наиболее подходящей пептидной последовательности и по меньшей мере 499 пептидных последовательностей-ловушек, и наивысший процент множества тестовых пептидных последовательностей прогнозируется в качестве презентируемых белком HLA, экспрессируемым в клетках посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA.
[0020] В некоторых вариантах осуществления машинно-обучаемая модель для прогнозирования презентации пептидов HLA имеет положительную прогностическую величину (PPV) по меньшей мере 0,1, когда информация об аминокислотах для множества тестовых пептидных последовательностей обрабатывается для получения множества тестовых прогнозов связывания, причем каждый тестовый прогноз связывания указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II, в клетке индивидуума связывает данную тестовую пептидную последовательность из множества тестовых пептидных последовательностей, где множество тестовых пептидных последовательностей содержит по меньшей мере 20 тестовых пептидных последовательностей, содержащих (i) по меньшей мере одну из наиболее подходящих пептидных последовательностей, идентифицированных масс-спектрометрией в качестве презентируемых белком HLA, экспрессируемым в клетках, и (ii) по меньшей мере 19 пептидных последовательностей-ловушек, содержащихся в белке, содержащем по меньшей мере одну пептидную последовательность, идентифицированную масс-спектрометрией в качестве презентируемой белком HLA, экспрессируемым в клетках, как например, одним белком HLA, экспрессируемым в клетках (например, моноаллельных клетках), где множество тестовых пептидных последовательностей включает соотношение 1:19 для по меньшей мере одной из наиболее подходящих пептидных последовательностей и по меньшей мере 19 пептидных последовательностей-ловушек и наивысший процент множества тестовых пептидных последовательностей прогнозируется в качестве связывающегося с белком HLA, экспрессируемых в клетках, посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA.
[0021] В некоторых вариантах осуществления между по меньшей мере одной из наиболее подходящих пептидных последовательностей и пептидными последовательностями-ловушками не существует перекрывания аминокислотной последовательности.
[0022] В некоторых вариантах осуществления машинно-обучаемая модель для прогнозирования презентации пептидов HLA имеет положительную прогностическую величину (PPV) по меньшей мере 0,08, 0,09, 0,1, 0,11, 0,12, 0,13, 0,14, 0,15, 0,16, 0,17, 0,18, 0,19, 0,2, 0,21, 0,22, 0,23, 0,24, 0,25, 0,26, 0,27, 0,28, 0,29, 0,3, 0,31, 0,32, 0,33, 0,34, 0,35, 0,36, 0,37, 0,38, 0,39, 0,4, 0,41, 0,42, 0,43, 0,44, 0,45, 0,46, 0,47, 0,48, 0,49, 0,5, 0,51, 0,52, 0,53, 0,54, 0,55, 0,56, 0,57, 0,58, 0,59, 0,6, 0,61, 0,62, 0,63, 0,64, 0,65, 0,66, 0,67, 0,68, 0,69, 0,7, 0,71, 0,72, 0,73, 0,74, 0,75, 0,76, 0,77, 0,78, 0,79, 0,8, 0,81, 0,82, 0,83, 0,84, 0,85, 0,86, 0,87, 0,88, 0,89, 0,9, 0,91, 0,92, 0,93, 0,94, 0,95, 0,96, 0,97, 0,98 или 0,99.
[0023] В некоторых вариантах осуществления по меньшей мере одна из наиболее подходящих пептидных последовательностей включает по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 или 100 наиболее подходящих пептидных последовательностей.
[0024] В некоторых вариантах осуществления по меньшей мере 499 пептидных последовательностей-ловушек включают по меньшей мере 500, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3200, 3300, 3400, 3500, 3600, 3700, 3800, 3900, 4000, 4100, 4200, 4300, 4400, 4500, 4600, 4700, 4800, 4900, 5000, 5100, 5200, 5300, 5400, 5500, 5600, 5700, 5800, 5900, 6000, 6100, 6200, 6300, 6400, 6500, 6600, 6700, 6800, 6900, 7000, 7100, 7200, 7300, 7400, 7500, 7600, 7700, 7800, 7900, 8000, 8100, 8200, 8300, 8400, 8500, 8600, 8700, 8800, 8900, 9000, 9100, 9200, 9300, 9400, 9500, 9600, 9700, 9800, 9900, 10000, 11000, 12000, 13000, 14000, 15000, 16000, 17000, 18000, 19000, 20000, 21000, 22000, 23000, 24000, 25000, 26000, 27000, 28000, 29000, 30000, 31000, 32000, 33000, 34000, 35000, 36000, 37000, 38000, 39000, 40000, 41000, 42000, 43000, 44000, 45000, 46000, 47000, 48000, 49000, 50000, 52500, 55000, 57500, 60000, 62500, 65000, 67500, 70000, 72500, 75000, 77500, 80000, 82500, 85000, 87500, 90000, 92500, 95000, 97500, 100000, 125000, 150000, 175000, 200000, 225000, 250000, 275000, 300000, 325000, 350000, 375000, 400000, 425000, 450000, 475000, 500000, 600000, 700000, 800000, 900000 или 1000000 пептидных последовательностей-ловушек. Специалист в данной области способен понять, что изменение соотношения наиболее подходящая последовательность: ловушка изменяет PPV.
[0025] В некоторых вариантах осуществления по меньшей мере 500 тестовых пептидных последовательностей включают по меньшей мере 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3200, 3300, 3400, 3500, 3600, 3700, 3800, 3900, 4000, 4100, 4200, 4300, 4400, 4500, 4600, 4700, 4800, 4900, 5000, 5100, 5200, 5300, 5400, 5500, 5600, 5700, 5800, 5900, 6000, 6100, 6200, 6300, 6400, 6500, 6600, 6700, 6800, 6900, 7000, 7100, 7200, 7300, 7400, 7500, 7600, 7700, 7800, 7900, 8000, 8100, 8200, 8300, 8400, 8500, 8600, 8700, 8800, 8900, 9000, 9100, 9200, 9300, 9400, 9500, 9600, 9700, 9800, 9900, 10000, 11000, 12000, 13000, 14000, 15000, 16000, 17000, 18000, 19000, 20000, 21000, 22000, 23000, 24000, 25000, 26000, 27000, 28000, 29000, 30000, 31000, 32000, 33000, 34000, 35000, 36000, 37000, 38000, 39000, 40000, 41000, 42000, 43000, 44000, 45000, 46000, 47000, 48000, 49000, 50000, 52500, 55000, 57500, 60000, 62500, 65000, 67500, 70000, 72500, 75000, 77500, 80000, 82500, 85000, 87500, 90000, 92500, 95000, 97500, 100000, 125000, 150000, 175000, 200000, 225000, 250000, 275000, 300000, 325000, 350000, 375000, 400000, 425000, 450000, 475000, 500000, 600000, 700000, 800000, 900000 или 1000000 тестовых пептидных последовательностей.
[0026] В некоторых вариантах осуществления наивысший процент представляет собой наивысшие 0,20%, 0,30%, 0,40%, 0,50%, 0,60%, 0,70%, 0,80%, 0,90%, 1,00%, 1,10%, 1,20%, 1,30%, 1,40%, 1,50%, 1,60%, 1,70%, 1,80%, 1,90%, 2,00%, 2,10%, 2,20%, 2,30%, 2,40%, 2,50%, 2,60%, 2,70%, 2,80%, 2,90%, 3,00%, 3,10%, 3,20%, 3,30%, 3,40%, 3,50%, 3,60%, 3,70%, 3,80%, 3,90%, 4,00%, 4,10%, 4,20%, 4,30%, 4,40%, 4,50%, 4,60%, 4,70%, 4,80%, 4,90%, 5,00%, 5,10%, 5,20%, 5,30%, 5,40%, 5,50%, 5,60%, 5,70%, 5,80%, 5,90%, 6,00%, 6,10%, 6,20%, 6,30%, 6,40%, 6,50%, 6,60%, 6,70%, 6,80%, 6,90%, 7,00%, 7,10%, 7,20%, 7,30%, 7,40%, 7,50%, 7,60%, 7,70%, 7,80%, 7,90%, 8,00%, 8,10%, 8,20%, 8,30%, 8,40%, 8,50%, 8,60%, 8,70%, 8,80%, 8,90%, 9,00%, 9,10%, 9,20%, 9,30%, 9,40%, 9,50%, 9,60%, 9,70%, 9,80%, 9,90%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20%.
[0027] В некоторых вариантах осуществления по меньшей мере одна наиболее подходящая пептидная последовательность включает по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 или 100 наиболее подходящих пептидных последовательностей.
[0028] В некоторых вариантах осуществления по меньшей мере 19 пептидных последовательностей-ловушек включают по меньшей мере 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200, 210, 220, 230, 240, 250, 260, 270, 280, 290, 300, 310, 320, 330, 340, 350, 360, 370, 380, 390, 400, 410, 420, 430, 440, 450, 460, 470, 480, 490, 500 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3200, 3300, 3400, 3500, 3600, 3700, 3800, 3900, 4000, 4100, 4200, 4300, 4400, 4500, 4600, 4700, 4800, 4900, 5000, 5100, 5200, 5300, 5400, 5500, 5600, 5700, 5800, 5900, 6000, 6100, 6200, 6300, 6400, 6500, 6600, 6700, 6800, 6900, 7000, 7100, 7200, 7300, 7400, 7500, 7600, 7700, 7800, 7900, 8000, 8100, 8200, 8300, 8400, 8500, 8600, 8700, 8800, 8900, 9000, 9100, 9200, 9300, 9400, 9500, 9600, 9700, 9800, 9900, 10000, 11000, 12000, 13000, 14000, 15000, 16000, 17000, 18000, 19000, 20000, 21000, 22000, 23000, 24000, 25000, 26000, 27000, 28000, 29000, 30000, 31000, 32000, 33000, 34000, 35000, 36000, 37000, 38000, 39000, 40000, 41000, 42000, 43000, 44000, 45000, 46000, 47000, 48000, 49000, 50000, 52500, 55000, 57500, 60000, 62500, 65000, 67500, 70000, 72500, 75000, 77500, 80000, 82500, 85000, 87500, 90000, 92500, 95000, 97500, 100000, 125000, 150000, 175000, 200000, 225000, 250000, 275000, 300000, 325000, 350000, 375000, 400000, 425000, 450000, 475000, 500000, 600000, 700000, 800000, 900000 или 1000000 пептидных последовательностей-ловушек.
[0029] В некоторых вариантах осуществления по меньшей мере 20 тестовых пептидных последовательностей включают по меньшей мере 500 тестовых пептидных последовательностей, включая по меньшей мере 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200, 210, 220, 230, 240, 250, 260, 270, 280, 290, 300, 310, 320, 330, 340, 350, 360, 370, 380, 390, 400, 410, 420, 430, 440, 450, 460, 470, 480, 490, 500 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3200, 3300, 3400, 3500, 3600, 3700, 3800, 3900, 4000, 4100, 4200, 4300, 4400, 4500, 4600, 4700, 4800, 4900, 5000, 5100, 5200, 5300, 5400, 5500, 5600, 5700, 5800, 5900, 6000, 6100, 6200, 6300, 6400, 6500, 6600, 6700, 6800, 6900, 7000, 7100, 7200, 7300, 7400, 7500, 7600, 7700, 7800, 7900, 8000, 8100, 8200, 8300, 8400, 8500, 8600, 8700, 8800, 8900, 9000, 9100, 9200, 9300, 9400, 9500, 9600, 9700, 9800, 9900, 10000, 11000, 12000, 13000, 14000, 15000, 16000, 17000, 18000, 19000, 20000, 21000, 22000, 23000, 24000, 25000, 26000, 27000, 28000, 29000, 30000, 31000, 32000, 33000, 34000, 35000, 36000, 37000, 38000, 39000, 40000, 41000, 42000, 43000, 44000, 45000, 46000, 47000, 48000, 49000, 50000, 52500, 55000, 57500, 60000, 62500, 65000, 67500, 70000, 72500, 75000, 77500, 80000, 82500, 85000, 87500, 90000, 92500, 95000, 97500, 100000, 125000, 150000, 175000, 200000, 225000, 250000, 275000, 300000, 325000, 350000, 375000, 400000, 425000, 450000, 475000, 500000, 600000, 700000, 800000, 900000 или 1000000 тестовых пептидных последовательностей.
[0030] В некоторых вариантах осуществления наивысший процент представляет собой наивысшие 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19%, 20%, 21%, 22%, 23%, 24%, 25%, 26%, 27%, 28%, 29%, 30%, 31%, 32%, 33%, 34%, 35%, 36%, 37%, 38%, 39% или 40%.
[0031] В некоторых вариантах осуществления PPV превышает соответствующий PPV в столбце 2 таблицы 11 для белка, кодируемого соответствующим аллелем HLA в таблице 11. В некоторых вариантах осуществления PPV по меньшей мере равен соответствующему PPV в столбце 3 таблицы 11 для белка, кодируемого соответствующим аллелем HLA в таблице 11.
[0032] В некоторых вариантах осуществления PPV равен или превышает соответствующий PPV в столбце 2 таблицы 12 для белка, кодируемого аллелем HLA класса II.
[0033] В некоторых вариантах осуществления PPV превышает соответствующий PPV в столбце 2 таблицы 16 для белка, кодируемого аллелем HLA класса II.
[0034] В некоторых вариантах осуществления индивидуумом является один индивидуум.
[0035] В некоторых вариантах осуществления индивидуумом является млекопитающее.
[0036] В некоторых вариантах осуществления индивидуумом является человек.
[0037] В некоторых вариантах осуществления клетки для обучения представляют собой клетки, экспрессирующие один белок, кодируемый аллелем HLA класса II, в клетке индивидуума.
[0038] В некоторых вариантах осуществления клетки для обучения представляют собой клетки с моноаллельным HLA, или клетки, экспрессирующие аллель HLA с аффинной меткой.
[0039] В некоторых вариантах осуществления клетка индивидуума включает злокачественные клетки.
[0040] В некоторых вариантах осуществления способ предназначен для идентификации пептидных последовательностей.
[0041] В некоторых вариантах осуществления способ предназначен для отбора пептидных последовательностей.
[0042] В некоторых вариантах осуществления способ предназначен для получения средства терапии злокачественной опухоли.
[0043] В некоторых вариантах осуществления способ предназначен для получения специфического для индивидуума средства терапии злокачественной опухоли.
[0044] В некоторых вариантах осуществления способ предназначен для получения специфического для злокачественных клеток средства терапии злокачественной опухоли.
[0045] В некоторых вариантах осуществления каждая пептидная последовательность из множества пептидных последовательностей ассоциирована со злокачественной опухолью.
[0046] В некоторых вариантах осуществления по меньшей мере одна пептидная последовательность из множества пептидных последовательностей сверхэкспрессируется злокачественной клеткой индивидуума.
[0047] В некоторых вариантах осуществления каждая пептидная последовательность из множества пептидных последовательностей сверхэкспрессируется злокачественной клеткой индивидуума.
[0048] В некоторых вариантах осуществления по меньшей мере одна пептидная последовательность из множества пептидных последовательностей представляет собой специфический для злокачественной клетки пептид.
[0049] В некоторых вариантах осуществления каждая пептидная последовательность из множества пептидных последовательностей представляет собой специфический для злокачественных клеток пептид.
[0050] В некоторых вариантах осуществления каждая пептидная последовательность из множества пептидных последовательностей экспрессируется злокачественной клеткой индивидуума.
[0051] В некоторых вариантах осуществления по меньшей мере одна пептидная последовательность из множества пептидных последовательностей не кодируется незлокачественной клеткой индивидуума.
[0052] В некоторых вариантах осуществления каждая пептидная последовательность из множества пептидных последовательностей не кодируется незлокачественной клеткой индивидуума.
[0053] В некоторых вариантах осуществления по меньшей мере одна пептидная последовательность из множества пептидных последовательностей не экспрессируется незлокачественной клеткой индивидуума.
[0054] В некоторых вариантах осуществления каждая пептидная последовательность из множества пептидных последовательностей не экспрессируется незлокачественной клеткой индивидуума.
[0055] В некоторых вариантах осуществления способ включает получение множества пептидных последовательностей индивидуума.
[0056] В некоторых вариантах осуществления способ включает получение множества полинуклеотидных последовательностей индивидуума.
[0057] В некоторых вариантах осуществления способ включает получение множества полинуклеотидных последовательностей индивидуума, которые кодируют множество пептидных последовательностей, кодируемых геномом или экзомом индивидуума, или патогеном или вирусом у индивидуума.
[0058] В некоторых вариантах осуществления способ включает получение множества полинуклеотидных последовательностей индивидуума, которые кодируют множество пептидных последовательностей, кодируемых геномом или экзомом индивидуума, процессором компьютера.
[0059] В некоторых вариантах осуществления способ включает получение множества полинуклеотидных последовательностей индивидуума посредством геномного или экзомного секвенирования.
[0060] В некоторых вариантах осуществления способ включает получение множества полинуклеотидных последовательностей индивидуума посредством полногеномного секвенирования или полноэкзомного секвенирования.
[0061] В некоторых вариантах осуществления обработка включает обработку посредством компьютерного процессора.
[0062] В некоторых вариантах осуществления обработка включает получение множества прогностических переменных на основе по меньшей мере информации об аминокислотах множества пептидных последовательностей.
[0063] В некоторых вариантах осуществления обработка множества прогностических переменных осуществляется с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA.
[0064] В некоторых вариантах осуществления этот один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, представляют собой один или более белков, кодируемых аллелем HLA класса II, которые экспрессируются у индивидуума.
[0065] В некоторых вариантах осуществления эти один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, представляют собой один или более белков, кодируемых аллелем HLA класса II, которые экспрессируются злокачественными клетками индивидуума.
[0066] В некоторых вариантах осуществления эти один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, представляют собой один белок, кодируемый аллелем HLA класса II клетки индивидуума.
[0067] В некоторых вариантах осуществления эти один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, представляют собой два, три, четыре, пять или шесть, или более белков, кодируемых аллелем HLA класса II клетки индивидуума.
[0068] В некоторых вариантах осуществления эти один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, в каждом случае представляют собой белок, кодируемый аллелем HLA класса II клетки индивидуума.
[0069] В некоторых вариантах осуществления способ дополнительно включает введение индивидууму композиции, содержащей одну или более из выбранных подгрупп пептидных последовательностей.
[0070] В некоторых вариантах осуществления идентификация множества пептидных последовательностей включает сравнение последовательностей ДНК, РНК или белков из злокачественных клеток индивидуума с последовательностями ДНК, РНК или белков из нормальных клеток индивидуума, где каждый из множества пептидов содержит по меньшей мере одну мутацию, которая присутствует в злокачественной клетке индивидуума и не присутствует в нормальной клетке индивидуума.
[0071] В некоторых вариантах осуществления машинно-обучаемая модель для прогнозирования презентации пептидов HLA включает множество прогностических переменных, идентифицированных по меньшей мере на основе обучающих данных, где обучающие данные включают информацию о пептидных последовательностях, включающую информацию об аминокислотных положениях, где обучающая информация о пептидных последовательностях ассоциирована с белком HLA, экспрессируемым к клетках; и функцию, отражающую взаимосвязь между информацией об аминокислотных положениях и вероятностью презентации, сгенерированную в качестве результата на основе информации об аминокислотных положениях и множества прогностических переменных.
[0072] В некоторых вариантах осуществления идентификация включает идентификацию на основе по меньшей мере множества прогнозов о презентации пептидной последовательности из множества пептидных последовательностей, которая имеет вероятность, превышающую пороговую величину вероятности прогноза презентации, презентироваться по меньшей мере одним из одного или более белков, кодируемых аллелем HLA класса II в клетке индивидуума.
[0073] В некоторых вариантах осуществления одна или более из 0,2% из множества тестовых пептидных последовательностей, спрогнозированных в качестве презентируемых машинной моделью прогнозирования для изучения презентации пептидов HLA, имеют вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентироваться по меньшей мере одним из одного или более белков, кодируемых аллелем HLA класса II в клетке индивидуума.
[0074] В некоторых вариантах осуществления каждые из 0,2% из множества тестовых пептидных последовательностей, спрогнозированных в качестве презентируемых машинной моделью прогнозирования для изучения презентации пептидов HLA, имеют вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентироваться по меньшей мере одним из одного или более белков, кодируемых аллелем HLA класса II в клетке индивидуума.
[0075] В некоторых вариантах осуществления количество положительных результатов ограничено величиной, равной количеству наиболее подходящих последовательностей.
[0076] В некоторых вариантах осуществления масс-спектрометрия представляет собой моноаллельную масс-спектрометрию.
[0077] В некоторых вариантах осуществления пептиды презентируются белком HLA, экспрессируемым в клетках посредством аутофагии.
[0078] В некоторых вариантах осуществления пептиды презентируются белком HLA, экспрессируемым в клетках посредством фагоцитоза.
[0079] В некоторых вариантах осуществления множество прогностических переменных включает прогностический параметр уровня экспрессии исходного белка, содержащего пептид.
[0080] В некоторых вариантах осуществления множество прогностических переменных включает прогностический параметр стабильности исходного белка, содержащего пептид.
[0081] В некоторых вариантах осуществления множество прогностических переменных включает прогностический параметр скорости деградации исходного белка, содержащего пептид.
[0082] В некоторых вариантах осуществления множество прогностических переменных включает прогностический параметр способности белка к расщеплению для исходного белка, содержащего пептид.
[0083] В некоторых вариантах осуществления множество прогностических переменных включает прогностический параметр клеточной или тканевой локализации исходного белка, содержащего пептид.
[0084] В некоторых вариантах осуществления множество прогностических переменных включает прогностический параметр способа внутриклеточного процессинга исходного белка, содержащего пептид, где способ процессинга исходного белка включает прогностический параметр того, подвергается ли белок-источник аутофагии, фагоцитозу и внутриклеточному транспорту, среди прочих.
[0085] В некоторых вариантах осуществления качество обучающих данных возрастает при использовании множества параметров качества.
[0086] В некоторых вариантах осуществления множество параметров качества включает общее устранение контаминирующих пептидов, высокую оцененную интенсивность пика, высокую оценку и высокую точность массы.
[0087] В некоторых вариантах осуществления оцененная интенсивность пика составляет по меньшей мере 50%.
[0088] В некоторых вариантах осуществления оцененная интенсивность пика составляет по меньшей мере 60%.
[0089] В некоторых вариантах осуществления оценка составляет по меньшей мере 7.
[0090] В некоторых вариантах осуществления точность массы составляет не более 5 м.д.
[0091] В некоторых вариантах осуществления пептиды, презентируемые белком HLA в клетках, представляют собой пептиды, презентируемые одним иммунопреципитированным белком HLA, экспрессируемым в клетках.
[0092] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, экспрессируемым в клетках, представляют собой пептиды, презентируемые одним экзогенным белком HLA, экспрессируемым в клетках.
[0093] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, экспрессируемым в клетках, представляют собой пептиды, презентируемые одним рекомбинантным белком HLA, экспрессируемым в клетках.
[0094] В некоторых вариантах осуществления множество прогностических переменных включает прогностическую переменную аффинности пептид-HLA.
[0095] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, включают пептиды, идентифицированные посредством поиска по отсутствию ферментной специфичности в базе данных пептидов без модификации.
[0096] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, включают пептиды, идентифицированные посредством поиска в базе данных пептидов с использованием стратегии обратного поиска в базе данных.
[0097] В некоторых вариантах осуществления белок HLA включает белок HLA-DR, HLA-DQ или HLA-DP.
[0098] В некоторых вариантах осуществления белок HLA включает белок HLA класса II, выбранный из группы, состоящей из: HLA-DPB1*01:01/HLA-DPA1*01:03, HLA-DPB1*02:01/HLA-DPA1*01:03, HLA-DPB1*03:01/HLA-DPA1*01:03, HLA-DPB1*04:01/HLA-DPA1*01:03, HLA-DPB1*04:02/HLA-DPA1*01:03, HLA-DPB1*06:01/HLA-DPA1*01:03,HLA-DQB1*02:01/HLA-DQA1*05:01, HLA-DQB1*02:02/HLA-DQA1*02:01, HLA-DQB1*06:02/HLA-DQA1*01:02, HLA-DQB1*06:04/HLA-DQA1*01:02, HLA-DRB1*01:01, HLA-DRB1*01:02, HLA-DRB1*03:01, HLA-DRB1*03:02, HLA-DRB1*04:01, HLA-DRB1*04:02, HLA-DRB1*04:03, HLA-DRB1*04:04, HLA-DRB1*04:05, HLA-DRB1*04:07, HLA-DRB1*07:01, HLA-DRB1*08:01, HLA-DRB1*08:02, HLA-DRB1*08:03, HLA-DRB1*08:04, HLA-DRB1*09:01, HLA-DRB1*10:01, HLA-DRB1*11:01, HLA-DRB1*11:02, HLA-DRB1*11:04, HLA-DRB1*12:01, HLA-DRB1*12:02, HLA-DRB1*13:01, HLA-DRB1*13:02, HLA-DRB1*13:03, HLA-DRB1*14:01, HLA-DRB1*15:01, HLA-DRB1*15:02, HLA-DRB1*15:03, HLA-DRB1*16:01, HLA-DRB3*01:01, HLA-DRB3*02:02, HLA-DRB3*03:01, HLA-DRB4*01:01, HLA-DRB5*01:01.
[0099] В некоторых вариантах осуществления HLA-DR является парным с DRA*01:01.
[0100] В некоторых вариантах осуществления белок HLA представляет собой белок HLA класса II, выбранный из группы, состоящей из: DPA*01:03/DPB*04:01, DRB1*01:01, DRB1*01:02, DRB1*03:01, DRB1*04:01, DRB1*04:02, DRB1*04:04, DRB1*04:05, DRB1*07:01, DRB1*08:01, DRB1*08:02, DRB1*08:03, DRB1*09:01, DRB1*11:01, DRB1*11:02, DRB1*11:04, DRB1*12:01, DRB1*13:01, DRB1*13:02, DRB1*13:03, DRB1*14:01, DRB1*15:01, DRB1*15:02, DRB1*15:03, DRB1*16:02, DRB3*01:01, DRB3*02:01, DRB3*02:02, DRB3*03:01, DRB4*01:01, DRB4*01:03 и DRB5*01:01.
[0101] В некоторых вариантах осуществления белок HLA-DR включает DRA*01:01 в форме димера.
[0102] В некоторых вариантах осуществления белок HLA включает белок HLA-DP, выбранный из группы, состоящей из: DPB1*01:01, DPB1*02:01, DPB1*02:02, DPB1*03:01, DPB1*04:01, DPB1*04:02, DPB1*05:01, DPB1*06:01, DPB1*11:01, DPB1*13:01, DPB1*17:01.
[0103] В некоторых вариантах осуществления белок HLA-DP является парным, включающим DPA1*01:03.
[0104] В некоторых вариантах осуществления белок HLA включает белковый комплекс HLA-DQ, выбранный из группы, состоящей из: A1*01:01+B1*05:01, A1*01:02+B1*06:02, A1*01:02+B1*06:04, A1*01:03+B1*06:03, A1*02:01+B1*02:02, A1*02:01+B1*03:03, A1*03:01+B1*03:02, A1*03:03+B1*03:01, A1*05:01+B1*02:01 и A1*05:05+B1*03:01.
[0105] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, включают пептиды, идентифицированные посредством сравнения спектров MS/MS HLA-пептидов со спектрами MS/MS одного или более пептидов или белков в базе данных пептидов или белков.
[0106] В некоторых вариантах осуществления мутация выбрана из группы, состоящей из точковой мутации, мутации участка сплайсинга, мутации со сдвигом рамки считывания, мутации со сквозным прочитыванием и мутации со слиянием генов.
[0107] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, имеют длину 15-40 аминокислот.
[0108] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, включают пептиды, идентифицированные посредством идентификации пептидов, презентируемых белком HLA, путем сравнения спектров MS/MS HLA-пептидов со спектрами MS/MS одного или более пептидов или белков в базе данных пептидов или белков.
[0109] В некоторых вариантах осуществления персонализированная терапия злокачественной опухоли дополнительно включает адъювант.
[0110] В некоторых вариантах осуществления персонализированная терапия злокачественной опухоли дополнительно включает ингибитор иммунной точки контроля.
[0111] В некоторых вариантах осуществления обучающие данные включают структурированные данные, данные временного ряда, неструктурированные данные, реляционные данные или любую их комбинацию.
[0112] В некоторых вариантах осуществления неструктурированные данные включают данные в виде изображений.
[0113] В некоторых вариантах осуществления реляционные данные включают данные потребительской системы, системы масштаба предприятия, операционной системы, веб-сайта, доступного через сеть прикладного программного интерфейса (API), или любой их комбинации.
[0114] В некоторых вариантах осуществления обучающие данные загружают в базу данных на основе облака.
[0115] В некоторых вариантах осуществления обучение проводят с использованием сверточных нейронных сетей.
[0116] В некоторых вариантах осуществления сверточные нейронные сети включают по меньшей мере два сверточных слоя.
[0117] В некоторых вариантах осуществления сверточные нейронные сети включают по меньшей мере одну стадию пакетной нормализации.
[0118] В некоторых вариантах осуществления сверточные нейронные сети включают по меньшей мере одну стадию пространственного исключения.
[0119] В некоторых вариантах осуществления сверточные нейронные сети включают по меньшей мере одну стадию глобального максимального пулинга.
[0120] В некоторых вариантах осуществления сверточные нейронные сети включают по меньшей мере один плотный слой.
[0121] В некоторых вариантах осуществления идентификация пептидных последовательности включает идентификацию пептидных последовательностей с мутацией, экспрессируемых в злокачественных клетках индивидуума.
[0122] В некоторых вариантах осуществления идентификация пептидных последовательностей включает идентификацию пептидных последовательностей, не экспрессируемых в нормальных клетках индивидуума.
[0123] В некоторых вариантах осуществления идентификация пептидных последовательностей включает идентификацию вирусных пептидных последовательностей.
[0124] В некоторых вариантах осуществления идентификация пептидных последовательностей включает идентификацию сверхэкспрессируемых пептидных последовательностей.
[0125] В рамках настоящего изобретения предусматривается способ идентификации пептидов, специфичных к HLA класса II, для иммунотерапии индивидуума, включающий: получение посредством компьютерного процессора пептида-кандидата, содержащего эпитоп, и множества пептидных последовательностей, каждая из которых содержит эпитоп; обработку компьютерным процессором информации об аминокислотах для множества пептидных последовательностей с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением прогноза презентации для каждой из множества пептидных последовательностей иммунной клетке, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II, может презентировать данную пептидную последовательность из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательности пептидов, презентируемых белком HLA, экспрессируемым в клетках и идентифицированных посредством масс-спектрометрии; отбор белка из одного или более белков, кодируемых аллелем HLA класса II клетки индивидуума, спрогнозированного в качестве связывающегося с пептидом-кандидатом посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA, где белок имеет вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентировать пептид-кандидат иммунной клетке; приведение пептида-кандидата в контакт с выбранным белком, так чтобы пептид-кандидат конкурировал с пептидом-местозаполнителем, ассоциированным с выбранным белком; и идентификацию пептида-кандидата в качестве пептида для иммунотерапии, специфичного к выбранному белку, на основе того, вытесняет ли пептид-кандидат местозаполнитель.
[0126] В некоторых вариантах осуществления получение включает идентификацию пептида-кандидата, где идентификация пептида-кандидата включает сравнение последовательностей ДНК, РНК или белков из злокачественных клеток с последовательностями ДНК, РНК или белков из нормальных клеток индивидуума.
[0127] В некоторых вариантах осуществления обработка включает идентификацию множества прогностических переменных на основе по меньшей мере информации об аминокислотах для множества пептидных последовательностей и обработку множества прогностических переменных с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA.
[0128] В некоторых вариантах осуществления машинно-обучаемая модель для прогнозирования презентации пептидов HLA включает множество прогностических переменных, идентифицированных по меньшей мере на основе обучающих данных, где обучающие данные включают: обучающую информацию о пептидных последовательностях, включающую информацию об аминокислотных положениях, где обучающая информация о пептидных последовательностях ассоциирована с белком HLA, экспрессируемым в клетках; и функцию, отражающую взаимосвязь между информацией об аминокислотных положениях и вероятностью презентации, сгенерированную в качестве результата на основе информации об аминокислотных положениях и множества прогностических переменных.
[0129] В некоторых вариантах осуществления количество положительных результатов ограничено величиной, равной количеству наиболее подходящих последовательностей.
[0130] В некоторых вариантах осуществления масс-спектрометрия представляет собой моноаллельную масс-спектрометрию.
[0131] В некоторых вариантах осуществления множество прогностических переменных включает любые один или более из следующих: прогностический параметр уровня экспрессии, прогностический параметр стабильности, прогностический параметр скорости деградации, прогностический параметр способности к расщеплению, прогностический параметр клеточной или тканевой локализации и прогностический параметр способа внутриклеточного процессинга, включающий прогностический параметр аутофагии, фагоцитоза и внутриклеточного транспорта, исходного белка, содержащего пептид.
[0132] В некоторых вариантах осуществления качество обучающих данных возрастает при использовании множества параметров качества.
[0133] В некоторых вариантах осуществления множество параметров качества включает общее устранение контаминирующих пептидов, высокую оцененную интенсивность пика, высокую оценку и высокую точность массы.
[0134] В некоторых вариантах осуществления оцененная интенсивность пика составляет по меньшей мере 50%.
[0135] В некоторых вариантах осуществления оцененная интенсивность пика составляет по меньшей мере 60%.
[0136] В некоторых вариантах осуществления пептид-местозаполнитель представляет собой пептид CLIP.
[0137] В некоторых вариантах осуществления пептид-местозаполнитель представляет собой пептид CMV.
[0138] В некоторых вариантах осуществления способ дополнительно включает измерение IC50 для вытеснения пептида-местозаполнителя пептидом-мишенью.
[0139] В некоторых вариантах осуществления IC50 для вытеснения пептида-местозаполнителя пептидом-мишенью составляет менее 500 нМ.
[0140] В некоторых вариантах осуществления по меньшей мере один белок из одного или более белков, кодируемых аллелем HLA класса II клетки индивидуума, представляет собой тетрамер или мультимер HLA класса II.
[0141] В некоторых вариантах осуществления пептид-мишень, далее идентифицируют посредством масс-спектрометрии.
[0142] В некоторых вариантах осуществления по меньшей мере один белок, кодируемый аллелем HLA класса II клетки индивидуума, представляет собой рекомбинантный белок.
[0143] В некоторых вариантах осуществления по меньшей мере один белок, кодируемый аллелем HLA класса II клетки индивидуума экспрессируется в эукариотической клетке.
[0144] В некоторых вариантах осуществления пептиды презентируются белком HLA, экспрессируемым в клетках посредством аутофагии.
[0145] В некоторых вариантах осуществления пептиды презентируются белком HLA, экспрессируемым в клетках посредством фагоцитоза.
[0146] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, экспрессируемым в клетках, представляют собой пептиды, презентируемые единичным иммунопреципитированным белком HLA, экспрессируемым в клетках.
[0147] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, экспрессируемым в клетках, представляют собой пептиды, презентируемые единичным экзогенным белком HLA, экспрессируемым в клетках.
[0148] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, экспрессируемым в клетках, представляют собой пептиды, презентируемые единичным рекомбинантным белком HLA, экспрессируемым в клетках.
[0149] В некоторых вариантах осуществления множество прогностических переменных включает прогностическую переменную аффинности пептид-HLA.
[0150] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, включают пептиды, идентифицированные по отсутствию ферментной специфичности в базе данных пептидов без модификации.
[0151] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, включают пептиды, идентифицированные посредством поиска в базе данных пептидов с использованием стратегии обратного поиска в базе данных.
[0152] В некоторых вариантах осуществления белок HLA включает белок HLA-DR, HLA-DQ или HLA-DP.
[0153] В некоторых вариантах осуществления иммунотерапия представляет собой иммунотерапию злокачественной опухоли.
[0154] В некоторых вариантах осуществления эпитоп представляет собой эпитоп, специфичный к злокачественной опухоли.
[0155] В некоторых вариантах осуществления по меньшей мере один белок, кодируемый аллелем HLA класса II, содержит по меньшей мере одну субъединицу альфа 1 и субъединицу бета 1 белка HLA, присутствующего в димерной форме.
[0156] В некоторых вариантах осуществления идентичность пептида известна.
[0157] В некоторых вариантах осуществления идентичность пептида неизвестна.
[0158] В некоторых вариантах осуществления идентичность пептида определяют посредством масс-спектрометрии.
[0159] В некоторых вариантах осуществления анализ обмена пептидами включает детекцию пептидных флуоресцентных зондов или меток.
[0160] В некоторых вариантах осуществления пептид-местозаполнитель представляет собой пептид CLIP. В некоторых вариантах осуществления пептид-местозаполнитель имеет аминокислотную последовательность PVSKMRMATPLLMQA (SEQ ID NO: 1).
[0161] В некоторых вариантах осуществления конструкция полинуклеиновой кислоты содержит экспрессирующий вектор, дополнительно содержащий один или более из: промотора, сигнала секреции, факторов димеризации, последовательности рибосомального пропуска, одной или более меток для очистки и/или детекции.
[0162] В некоторых вариантах осуществления последовательность пептида-местозаполнителя кодируется последовательностью нуклеиновой кислоты в векторе.
[0163] В некоторых вариантах осуществления последовательность, кодирующая расщепляемый домен, находится между последовательностью, кодирующей пептид-местозаполнитель, и пептидом HLA-бета 1.
[0164] В рамках настоящего изобретения предусматривается способ анализа иммуногенности пептида, связывающегося с MHC класса II, включающий: выбор белка, кодируемого аллелем HLA класса II, спрогнозированным посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA в качестве пептида, связывающего MHC класса II, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA организована для генерирования прогноза презентации для данной пептидной последовательности, причем прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II, могут презентировать данную пептидную последовательность, и где белок имеет вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентировать пептид, связывающийся с MHC класса II; приведение пептида в контакт с выбранным белком, так чтобы пептид конкурировал с пептидом-местозаполнителем, ассоциированным с выбранным белком, и вытеснял пептид-местозаполнитель, тем самым образуя комплекс, содержащий белок HLA класса II и пептид, связывающий MHC класса II; приведение комплекса в контакт с CD4+ T-клеткой и анализ в отношении одного или более параметров активации CD4+ T-клеток, выбранных из группы, состоящей из: индукции цитокина, индукции хемокина и экспрессии маркера клеточной поверхности.
[0165] В некоторых вариантах осуществления аллель HLA класса II представляет собой тетрамер или мультимер.
[0166] В некоторых вариантах осуществления цитокин представляет собой IL-2.
[0167] В рамках настоящего изобретения предусматривается способ индукции активации CD4+ T-клеток у индивидуума для иммунотерапии злокачественной опухоли, причем способ включает: идентификацию пептидной последовательности, ассоциированной со злокачественной опухолью и содержащей мутацию злокачественной опухоли, где идентификация пептидной последовательности включает сравнение последовательностей ДНК, РНК или белков из злокачественных клеток индивидуума с последовательностями ДНК, РНК или белков из нормальных клеток индивидуума; выбор белка, кодируемого аллелем HLA класса II, который в норме экспрессируется клеткой индивидуума и спрогнозирован посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA в качестве связывающегося с пептидом; где модель прогнозирования имеет положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва по меньшей мере 0,1%, 0,1%-50% или не более 50% и где белок имеет вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентировать идентифицированную пептидную последовательность; приведение идентифицированного пептида в контакт с выбранным белком, кодируемым аллелем HLA класса II, для подтверждения того, конкурирует ли идентифицированный пептид с пептидом-местозаполнителем, связанным с выбранным белком, кодируемым аллелем HLA класса II, вытесняя пептид-местозаполнитель с величиной IC50 менее 500 нМ; необязательно, очистку идентифицированного пептида; и введение эффективного количества полипептида, содержащего последовательность идентифицированного пептида или полинуклеотида, кодирующего полипептид, индивидууму.
[0168] В рамках настоящего изобретения предусматривается способ скрининга лекарственных средств, содержащих полипептидную последовательность, в отношении иммуногенности у индивидуума, включающий: получение посредством компьютерного процессора множества пептидных последовательностей полипептидной последовательности; обработку посредством компьютерного процессора информации об аминокислотах для множества пептидных последовательностей с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA, с получением прогноза презентации для каждой из множества пептидных последовательностей, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем MHC класса I или II клетки индивидуума, может презентировать последовательность эпитопа данной пептидной последовательности из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательности, ассоциированную с экспрессией белка HLA в клетках; определение или прогнозирование того, что каждая из множества пептидных последовательностей полипептидной последовательности не будет иммуногенной для индивидуума, на основе множества прогнозов презентации; и введение индивидууму композиции, содержащей лекарственное средство.
[0169] В рамках настоящего изобретения предусматривается способ получения тетрамеров или мультимеров HLA класса II посредством конъюгации четырех индивидуальных гетеродимеров альфа 1 и бета 1 белка HLA, причем способ включает: экспрессию в эукариотической клетке вектора, содержащего последовательность нуклеиновой кислоты, кодирующую альфа-цепь и бета-цепь белка HLA, сигнал секреции, мотив биотинилирования и по меньшей мере одну метку для идентификации или для очистки, так чтобы каждый гетеродимер альфа 1 и бета 1 белка HLA секретировался в димеризованном состоянии, где гетеродимер связан с пептидом-местозаполнителем, очистку секретированного гетеродимера из клеточной среды, подтверждение активности связывания пептида с использованием анализа обмена пептидами, добавление стрептавидина, тем самым конъюгируя гетеродимеры в тетрамеры, очистку тетрамеров и получение выхода более 1 мг/л. Аналогично, также можно получать мультимеры, например, пентамеры, гексамеры или октамеры, которые в равной степени предусматриваются в рамках настоящего изобретения.
[0170] В некоторых вариантах осуществления вектор содержит промотор CMV.
[0171] В некоторых вариантах осуществления вектор содержит последовательность, кодирующую пептид-местозаполнитель, связанный через расщепляемый участок с цепью бета 1.
[0172] В некоторых вариантах осуществления анализ обмена пептидами вовлекает предшествующее отщепление пептида-местозаполнителя от бета-цепи.
[0173] В некоторых вариантах осуществления расщепляемый участок представляет собой участок расщепления тромбином.
[0174] В некоторых вариантах осуществления анализ обмена пептидами представляет собой анализ FRET.
[0175] В некоторых вариантах осуществления очистку проводят посредством любой из: колоночной хроматографии, ионообменной хроматографии, эксклюзионной хроматографии, аффинной хроматографии или LC-MS.
[0176] в рамках настоящего изобретения предусматривается тетрамер или мультимер HLA класса II, содержащий гетеродимеры либо HLA-DR, либо HLA-DP, либо HLA-DQ, причем каждый гетеродимер содержит цепь альфа и бета, где гетеродимер очищен и присутствует в концентрации более 1 мг/л.
[0177] В некоторых вариантах осуществления тетрамеры HLA класса II выбраны из таблиц 8A-8C.
[0178] В некоторых вариантах осуществления тетрамер HLA класса II содержит гетеродимерные пары, выбранные из группы, состоящей из: белков HLA-DR, HLA-DP и HLA-DQ.
[0179] В некоторых вариантах осуществления белок HLA представляет собой белок HLA класса II, выбранный из группы, состоящей из: HLA-DPB1*01:01/HLA-DPA1*01:03, HLA-DPB1*02:01/HLA-DPA1*01:03, HLA-DPB1*03:01/HLA-DPA1*01:03, HLA-DPB1*04:01/HLA-DPA1*01:03, HLA-DPB1*04:02/HLA-DPA1*01:03, HLA-DPB1*06:01/HLA-DPA1*01:03, HLA-DQB1*02:01/HLA-DQA1*05:01,HLA-DQB1*02:02/HLA-DQA1*02:01, HLA-DQB1*06:02/HLA-DQA1*01:02,HLA-DQB1*06:04/HLA-DQA1*01:02, HLA-DRB1*01:01, HLA-DRB1*01:02, HLA-DRB1*03:01, HLA-DRB1*03:02, HLA-DRB1*04:01, HLA-DRB1*04:02, HLA-DRB1*04:03, HLA-DRB1*04:04, HLA-DRB1*04:05, HLA-DRB1*04:07, HLA-DRB1*07:01, HLA-DRB1*08:01, HLA-DRB1*08:02, HLA-DRB1*08:03, HLA-DRB1*08:04, HLA-DRB1*09:01, HLA-DRB1*10:01, HLA-DRB1*11:01, HLA-DRB1*11:02, HLA-DRB1*11:04, HLA-DRB1*12:01, HLA-DRB1*12:02, HLA-DRB1*13:01, HLA-DRB1*13:02, HLA-DRB1*13:03, HLA-DRB1*14:01, HLA-DRB1*15:01, HLA-DRB1*15:02, HLA-DRB1*15:03, HLA-DRB1*16:01, HLA-DRB3*01:01, HLA-DRB3*02:02, HLA-DRB3*03:01, HLA-DRB4*01:01 и HLA-DRB5*01:01.
[0180] В некоторых вариантах осуществления гетеродимерная пара экспрессируется в эукариотической клетке.
[0181] В некоторых вариантах осуществления гетеродимерные пары кодируются вектором.
[0182] В рамках настоящего изобретения предусматривается вектор, где вектор содержит последовательность нуклеиновой кислоты, кодирующую альфа-цепь и бета-цепь белка HLA, описанного в настоящем описании, секреторный сигнал, мотив биотинилирования и по меньшей мере одну метку для идентификации или для очистки, так чтобы каждый из гетродимеров альфа 1 и бета 1 белков HLA секретировался в димеризованном состоянии, где секретируемый гетеродимер необязательно связан с пептидом-местозаполнителем.
[0183] В рамках настоящего изобретения предусматривается клетка, содержащая вектор, описанный в настоящем описании.
[0184] В некоторых вариантах осуществления гетеродимеры HLA класса II секретируются из эукариотических клеток в среду для культивирования клеток, которая далее очищена посредством любой из: колоночной хроматографии, ионообменной хроматографии, эксклюзионной хроматографии, аффинной хроматографии или LC-MS.
[0185] В рамках настоящего изобретения предусматривается способ скрининга лекарственных средств, содержащих полипептидную последовательность, в отношении иммуногенности у индивидуума, включающий: получение посредством компьютерного процессора множества пептидных последовательностей полипептидной последовательности; обработку посредством компьютерного процессора информации об аминокислотах для множества пептидных последовательностей с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением прогноза презентации для каждой из множества пептидных последовательностей, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем MHC класса I или II клетки индивидуума, может презентировать последовательность эпитопа данной пептидной последовательности из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательности пептидов, презентируемых белком HLA, экспрессируемым в клетках и идентифицированных масс-спектрометрией; и определение или прогнозирование того, что по меньшей мере одна из множества пептидных последовательностей полипептидной последовательности будет иммуногенной у индивидуума, на основе множества прогнозов презентации.
[0186] В рамках настоящего изобретения предусматривается способ скрининга лекарственного средства, содержащего полипептидную последовательность, в отношении иммуногенности у индивидуума, причем способ включает: ввод информации об аминокислотах для пептидных последовательностей полипептидной последовательности с использованием компьютерного процессора в машинно-обучаемую модель для прогнозирования презентации пептидов HLA с получением набора прогнозов презентации для пептидных последовательностей, причем каждый прогноз презентации отражает вероятность того, что один или более белков, кодируемых аллелем MHC класса I или II клетки индивидуума, будут презентировать последовательность эпитопа данной пептидной последовательности; где машинно-обучаемая модель для прогнозирования презентации пептидов HLA включает: множество прогностических переменных, идентифицированных по меньшей мере на основе обучающих данных, где обучающие данные включают: информацию о последовательности пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; обучающую информацию о пептидных последовательностях, включающую информацию об аминокислотных положениях, где обучающая информация о пептидных последовательностях ассоциирована с белком HLA, экспрессируемым в клетках; и функцию, отражающую взаимосвязь между информацией об аминокислотных положениях, полученной в качестве вводной информации, и вероятностью презентации, сгенерированную в качестве результата на основе информации об аминокислотных положениях и прогностических переменных; определение или прогнозирование того, что каждая из пептидных последовательностей полипептидной последовательности не будет иммуногенной для индивидуума на основе набора прогнозов презентации; и введение индивидууму композиции, содержащей лекарственное средство.
[0187] В рамках настоящего изобретения предусматривается способ скрининга лекарственного средства, содержащего полипептидную последовательность, в отношении иммуногенности у индивидуума, причем способ включает: ввод информации об аминокислотах для пептидных последовательностей полипептидной последовательности с использованием компьютерного процессора в машинно-обучаемую модель для прогнозирования презентации пептидов HLA с получением набора прогнозов презентации для пептидных последовательностей, причем каждый прогноз презентации отражает вероятность того, что один или более белков, кодируемых аллелем MHC класса I или II клетки индивидуума, будут презентировать последовательность эпитопа данной пептидной последовательности; где машинно-обучаемая модель для прогнозирования презентации пептидов HLA включает: множество прогностических переменных, идентифицированных по меньшей мере на основе обучающих данных; где обучающие данные включают: информацию о последовательности пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; обучающую информация о пептидных последовательностях, включающую информацию об аминокислотных положениях, где обучающая информация о пептидных последовательностях ассоциирована с белком HLA, экспрессируемым в клетках; и функцию, отражающую взаимосвязь между информацией об аминокислотных положениях, полученной в качестве вводной информации, и вероятностью презентации, сгенерированную в качестве результата на основе информации об аминокислотных положениях и прогностических переменных; определение или прогнозирование того, что по меньшей мере одна из пептидных последовательностей полипептида будет иммуногенной для индивидуума на основе прогнозов презентации.
[0188] В рамках настоящего изобретения предусматривается способ скрининга лекарственного средства, содержащего полипептидную последовательность, в отношении иммуногенности у индивидуума, включающий: получение посредством компьютерного процессора множества пептидных последовательностей полипептидной последовательности; обработку посредством компьютерного процессора информации об аминокислотах для множества пептидных последовательностей с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением прогноза презентации для каждой из множества пептидных последовательностей, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем MHC класса I или II клетки индивидуума, может презентировать последовательность эпитопа данной пептидной последовательности из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательности, ассоциированную с белком HLA, экспрессируемым в клетках; определение или прогнозирование того, что каждая из множества пептидных последовательностей полипептидной последовательности не будет иммуногенной для индивидуума на основе множества прогнозов презентации; и введение индивидууму композиции, содержащей лекарственное средство.
[0189] В некоторых вариантах осуществления способ дополнительно включает решение не вводить лекарственное средство индивидууму.
[0190] В некоторых вариантах осуществления лекарственное средство содержит антитело или его связывающий фрагмент.
[0191] В некоторых вариантах осуществления пептидные последовательности полипептидной последовательности имеют длину 8, 9, 10, 11 или 12 аминокислот, и где белок, кодируемый аллелем MHC класса I или II клетки индивидуума, представляет собой белок, кодируемый аллелем MHC класса I клетки индивидуума.
[0192] В некоторых вариантах осуществления пептидные последовательности полипептидной последовательности имеют длину 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25 аминокислот, и где белок, кодируемый аллелем MHC класса I или II клетки индивидуума, представляет собой белок, кодируемый аллелем MHC класса II клетки индивидуума.
[0193] В рамках настоящего изобретения предусматривается способ лечения индивидуума с аутоиммунным заболеванием или состоянием, включающий: (a) идентификацию или прогнозирование эпитопа экспессируемого белка, презентируемого MHC класса I или II клетки индивидуума, где на комплекс, содержащий идентифицированный или спрогнозированный эпитоп и MHC класса I или II, нацелена CD8 или CD4 T-клетка индивидуума; (b) идентификацию T-клеточного рецептора (TCR), который связывается с комплексом; (c) экспрессию TCR в регуляторной T-клетке индивидуума или аллогенной регуляторной T-клетке; и (d) введение регуляторной T-клетки, экспрессирующей TCR, индивидууму.
[0194] В некоторых вариантах осуществления аутоиммунное заболевание или состояние представляет собой диабет.
[0195] В некоторых вариантах осуществления клетка представляет собой островковую клетку.
[0196] В рамках настоящего изобретения предусматривается способ лечения индивидуума с аутоиммунным заболеванием или состоянием, включающий введение индивидууму регуляторной T-клетки, экспрессирующей T-клеточный рецептор (TCR), который связывается с комплексом, содержащим: (i) эпитоп экспрессируемого белка, идентифицированного или спрогнозированного в качестве презентируемого MHC класса I или II клетки индивидуума, и (ii) MHC класса I или II, где на комплекс нацеливается CD8 или CD4 T-клетка индивидуума.
[0197] В рамках настоящего изобретения предусматривается компьютерная система для идентификации пептидных последовательностей для персонализированной терапии злокачественной опухоли у индивидуума, включающая: базу данных, которая организована для хранения множества пептидных последовательностей индивидуума; и один или более компьютерных процессоров, функционально сопряженных с указанной базой данных, где указанные один или более компьютерных процессоров индивидуально коллективно запрограммированы для: обработки информации об аминокислотах для множества пептидных последовательностей с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением прогноза презентации для каждой из множества пептидных последовательностей, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем MHC класса II клетки индивидуума, могут презентировать данную пептидную последовательность из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательности пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; и отбора подгруппы из множества пептидных последовательностей для персонализированной терапии злокачественной опухоли у индивидуума на основе по меньшей мере множества прогнозов презентации.
[0198] В рамках настоящего изобретения предусматривается компьютерная система для идентификации пептидов, специфичных к HLA класса II, для иммунотерапии индивидуума, включающая: базу данных, которая организована для хранения последовательности пептида-кандидата, содержащего эпитоп, и множества пептидных последовательностей, каждая из которых содержит эпитоп; и один или более компьютерных процессоров, функционально сопряженных с указанной базой данных, где указанные один или более компьютерных процессоров индивидуально коллективно запрограммированы для: обработки информации об аминокислотах для множества пептидных последовательностей с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением прогноза презентации для каждой из множества пептидных последовательностей иммунной клетке, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II, могут презентировать данную пептидную последовательность из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательности пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; выбора белка из одного или более белков, кодируемых аллелем HLA класса II клетки индивидуума, спрогнозированных в качестве связывающихся с пептидом-кандидатом посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA, где белок имеет вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентировать пептид-кандидат иммунной клетке; и идентификации пептида-кандидата в качестве пептида для иммунотерапии, специфической в отношении выбранного белка, на основе того, вытесняет ли пептид-кандидат пептид-местозаполнитель, при контакте пептида-кандидата с выбранном белком, так чтобы пептид-кандидат конкурировал с пептидом-местозаполнителем, связанным с выбранным белком.
[0199] В рамках настоящего изобретения предусматривается компьютерная система для скрининга лекарственных средств, содержащая полипептидную последовательность, в отношении иммуногенности у индивидуума, содержащая: базу данных, которая организована для хранения множества пептидных последовательностей полипептидной последовательности; и один или более компьютерных процессоров, функционально сопряженных с указанной базой данных, где указанные один или более компьютерных процессоров индивидуально коллективно запрограммированы для: обработки информации об аминокислотах для множества пептидных последовательностей с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением прогноза презентации для каждой из множества пептидных последовательностей, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем MHC класса I или II клетки индивидуума, могут презентировать последовательность эпитопа данной пептидной последовательности из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательности, ассоциированную с белком HLA, экспрессируемым в клетке; и определения или прогнозирования того, что каждая из множества пептидных последовательностей полипептидной последовательность не будет иммуногенной для индивидуума, на основе множества прогнозов презентации, где композицию, содержащую лекарственное средство, вводят индивидууму.
[0200] В рамках настоящего изобретения предусматривается компьютерная система для скрининга лекарственных средств, содержащая полипептидную последовательность, в отношении иммуногенности у индивидуума, содержащая: базу данных, которая организована для хранения множества пептидных последовательностей полипептидной последовательности; и один или более компьютерных процессоров, функционально сопряженных с указанной базой данных, где указанные один или более компьютерных процессоров индивидуально коллективно запрограммированы для: обработки информации об аминокислотах для множества пептидных последовательностей с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением прогноза презентации для каждой из множества пептидных последовательностей, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем MHC класса I или II клетки индивидуума, может презентировать последовательность эпитопа данной пептидной последовательности из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательности пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; и определения или прогнозирования того, что по меньшей мере одна из множества пептидных последовательностей полипептидной последовательности будет иммуногенной у индивидуума, на основе множества прогнозов презентации.
[0201] В рамках настоящего изобретения предусматривается машинно-читаемый носитель для долговременного хранения информации, содержащий машинно-выполняемый код, который при выполнении посредством одного или более компьютерных процессоров выполняет способ идентификации пептидных последовательностей для персонализированной терапии злокачественной опухоли у индивидуума, причем указанный способ включает: получение множества пептидных последовательностей индивидуума; обработку информации об аминокислотах для множества пептидных последовательностей с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением прогноза презентации для каждой из множества пептидных последовательностей, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем MHC класса II клетки индивидуума, может презентировать данную пептидную последовательность из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательностях пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; и выбора подгруппы из множества пептидных последовательностей для персонализированной терапии злокачественной опухоли индивидуума на основе по меньшей мере множества прогнозов презентации.
[0202] В рамках настоящего изобретения предусматривается машинно-читаемый носитель для долговременного хранения информации, содержащий машинно-выполняемый код, который при выполнении посредством одного или более компьютерных процессоров выполняет способ идентификации пептидов, специфичных к HLA класса II, для иммунотерапии индивидуума, включающий: получение пептида-кандидата, содержащего эпитоп и множества пептидных последовательности, каждая из которых содержит эпитоп; обработку информации об аминокислотах из множества пептидных последовательностей посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением прогноза презентации каждой из множества пептидных последовательностей иммунной клетке, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II, может презентировать данную пептидную последовательность из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательностях пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; отбор белка из одного или более белков, кодируемых аллелем HLA класса II клетки индивидуума, спрогнозированных в качестве связывающихся в пептидом-кандидатом посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA, где белок имеет вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентировать пептид-кандидат иммунной клетке; и идентификацию пептида-кандидата в качестве пептида иммунотерапии, специфичного к отобранному белку, на основе того, вытесняет ли пептид-кандидат пептид-местозаполнитель, при приведении пептида в контакт с выбранным белком, так чтобы пептид-кандидат с пептидом-местозаполнителем.
[0203] В рамках настоящего изобретения предусматривается машинно-читаемый носитель для долговременного хранения информации, содержащий машинно-выполняемый код, который при выполнении посредством одного или более компьютерных процессоров выполняет способ скрининга лекарственного средства, содержащего полипептидную последовательность, в отношении иммуногенности у индивидуума, включающий: получение множества пептидных последовательностей полипептидной последовательности; обработку информации об аминокислотах из множества пептидных последовательностей с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением прогноза презентации для каждой из множества пептидных последовательностей, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем MHC класса I или II клетки индивидуума, может презентировать последовательность эпитопа данной пептидной последовательности из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательности, ассоциированную с белком HLA, экспрессируемым в клетках; и определение или прогнозирование того, что каждая из множества пептидных последовательностей полипептидной последовательности не будет иммуногенной для индивидуума, на основе множества прогнозов презентации, где композицию, содержащую лекарственное средство, вводят индивидууму.
[0204] В рамках настоящего изобретения предусматривается машинно-читаемый носитель для долговременного хранения информации, содержащий машинно-выполняемый код, который при выполнении посредством одного или более компьютерных процессоров выполняет способ скрининга лекарственного средства, содержащего полипептидную последовательность, в отношении иммуногенности у индивидуума, включающий: получение множества пептидных последовательностей полипептидной последовательности; обработку информации об аминокислотах из множества пептидных последовательностей с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением прогноза презентации для каждой из множества пептидных последовательностей, причем каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем MHC класса I или II клетки индивидуума, может презентировать последовательность эпитопа данной пептидной последовательности из множества пептидных последовательностей, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательностях пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; и определение или прогнозирование того, что по меньшей мере один из множества пептидных последовательностей полипептидной последовательности будет иммуногенным у индивидуума, на основе множества прогнозов презентации.
[0205] В рамках настоящего изобретения предусматривается способ включающий: обработку информации об аминокислотах для множества последовательностей пептидов-кандидатов с использованием машинно-обучаемой модели для прогнозирования презентации пептидов HLA с получением множества прогнозов презентации, где каждая из последовательностей пептидов-кандидатов в множестве кодируется геномом или экзомом индивидуума, где множество прогнозов презентации включает прогноз презентации HLA для каждой из множества пептидных последовательностей-кандидатов, где каждый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, может презентировать данную пептидную последовательность-кандидат, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательностях обучающих пептидов, идентифицированных посредством масс-спектрометрии в качестве презентируемых белком HLA, экспрессируемым в обучающих клетках; и идентификацию, на основе по меньшей мере множества прогнозов презентации, пептидной последовательности из множества пептидных последовательностей, которая имеет вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентироваться по меньшей мере одним из одного или более белков, кодируемых аллелем HLA класса II в клетке индивидуума; где машинно-обучаемая модель для прогнозирования презентации пептидов HLA имеет положительную прогностическую величину (PPV) по меньшей мере 0,07, когда информация об аминокислотах для множества тестовых пептидных последовательности обрабатывается с получением множества тестовых прогнозов презентации, причем каждый тестовый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, могут презентировать данную тестовую пептидную последовательность из множества тестовых пептидных последовательностей, где множество тестовых пептидных последовательностей содержит по меньшей мере 500 тестовых пептидных последовательностей, содержащих (i) по меньшей мере одну наиболее подходящую пептидную последовательность, идентифицированную посредством масс-спектрометрии в качестве презентируемой белком HLA, экспрессируемым в клетках, и (ii) по меньшей мере 499 последовательностей пептидов-ловушек, находящихся в белке, кодируемом геномом организма, где организм и индивидуум принадлежат к одному виду, где множество тестовых пептидных последовательностей имеет соотношение 1:499 по меньшей мере одной из наиболее подходящих пептидных последовательностей и по меньшей мере 499 пептидных последовательностей-ловушек и 0,2% от множества тестовых пептидных последовательностей прогнозируется в качестве презентируемых белком HLA, экспрессируемым в клетках, посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA.
[0206] В рамках настоящего изобретения предусматривается способ, включающий: обработку информации об аминокислотах для множества пептидных последовательностей, кодируемых геномом или экзомом индивидуума, с использованием машинно-обучаемой модели для прогнозирования связывания пептидов HLA с получением множества прогнозов связывания, где множество прогнозов связывания включает прогноз связывания HLA для каждой из множества пептидных последовательностей-кандидатов, причем каждый из прогнозов связывания указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, связывается с данной последовательностью пептида-кандидата из множества последовательностей пептидов-кандидатов, где машинно-обучаемая модель для прогнозирования связывания пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательности пептидов, идентифицированных в качестве связывающихся с белком HLA класса II или с аналогом белка HLA класса II; и идентификацию, на основе по меньшей мере множества прогнозов связывания, пептидной последовательность из множества пептидных последовательностей, которая имеет вероятность, превышающую пороговую величину вероятности прогноза связывания, которая имеет вероятность, превышающую пороговую величину вероятности для прогноза связывания, связываться по меньшей мере с одним из одного или более белков, кодируемых аллелем HLA класса II в клетке индивидуума; где машинно-обучаемая модель для прогнозирования связывания пептидов HLA имеет положительную прогностическую величину (PPV) по меньшей мере 0,1, когда информация об аминокислотах для множества тестовых пептидных последовательностей обрабатывается для получения множества тестовых прогнозов связывания, причем каждый тестовый прогноз связывания указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, связывается с данной тестовой пептидной последовательностью из множества тестовых пептидных последовательностей, где множество тестовых пептидных последовательностей содержит по меньшей мере 50 тестовых пептидных последовательностей, содержащих (i) по меньшей мере одну наиболее подходящую пептидную последовательность, идентифицированную посредством масс-спектрометрии в качестве презентируемой белком HLA, экспрессируемым в клетках, и (ii) по меньшей мере 19 последовательностей пептидов-ловушек, содержащихся в белке, содержащем пептидную последовательность, идентифицированную посредством масс-спектрометрии в качестве презентируемой белком HLA, экспрессируемым в клетках, где организм и индивидуум принадлежат к одному виду, где множество тестовых пептидных последовательностей содержит соотношение 1:19 по меньшей мере одной наиболее подходящей пептидной последовательности и по меньшей мере 19 последовательностей пептидов-ловушек и прогнозируется, и 5% от множества тестовых пептидных последовательностей связываются с белком HLA, экспрессируемым в клетках, посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA.
[0207] В некоторых вариантах осуществления машинно-обучаемая модель для прогнозирования презентации пептидов HLA обучена с использованием обучающих данных, включающих информацию о последовательностях обучающих пептидов, идентифицированных посредством масс-спектрометрии в качестве презентируемых белком HLA, экспрессируемым в обучающих клетках.
[0208] В некоторых вариантах осуществления одна или более из 0,2% из множества тестовых пептидных последовательностей, спрогнозированных в качестве презентируемых посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA, имеют вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентироваться по меньшей мере одним из одного или более белков, кодируемых аллелем HLA класса II, в клетке индивидуума.
[0209] В некоторых вариантах осуществления каждая из 0,2% из множества тестовых пептидных последовательностей, спрогнозированных в качестве презентируемых посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA, имеют вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентироваться по меньшей мере одним из одного или более белков, кодируемых аллелем HLA класса II, в клетке индивидуума.
[0210] В некоторых вариантах осуществления PPV превышает соответствующую PPV в столбце 2 таблицы 11 для белка, кодируемого соответствующим аллелем HLA в таблице 13. В некоторых вариантах осуществления PPV по меньшей мере равна соответствующей PPV в столбце 3 таблицы 11 для белка, кодируемого соответствующим аллелем HLA в таблице 11.
[0211] В некоторых вариантах осуществления PPV превышает соответствующую PPV в столбце 2 таблицы 12 для белка, кодируемого аллелем HLA класса II.
[0212] В некоторых вариантах осуществления PPV по меньшей мере равна соответствующей PPV в столбце 2 таблицы 16 для белка, кодируемого соответствующим аллелем HLA в таблице 16.
[0213] В рамках настоящего изобретения предусматривается способ получения средства персонализированной терапии злокачественной опухоли, причем способ включает: идентификацию пептидных последовательностей, где пептидные последовательности ассоциированы со злокачественной опухолью, где идентификация включает сравнение последовательностей ДНК, РНК или белков из злокачественных клеток индивидуума с последовательностями ДНК, РНК или белков из нормальных клеток индивидуума; ввод информации об аминокислотных положениях идентифицированных пептидных последовательностей с использованием компьютерного процессора в машинно-обучаемую модель для прогнозирования презентации пептидов HLA с получением набора прогнозов презентации для идентифицированных пептидных последовательностей, причем каждый прогноз презентации отражает вероятность того, что один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, будет презентировать данную идентифицированную пептидную последовательность; где машинно-обучаемая модель для прогнозирования презентации пептидов HLA включает: множество прогностических переменных, идентифицированных по меньшей мере на основе обучающих данных, где обучающие данные включают: информацию о последовательностях пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; обучающую информацию о пептидных последовательностях, включающую информацию об аминокислотных положениях, где обучающая информация о пептидных последовательностях ассоциирована с белком HLA, экспрессируемым в клетках; и функцию, отражающую взаимосвязь между информацией об аминокислотных положениях, получаемую в качестве вводной информации, и вероятностью презентации, сгенерированную в качестве результата на основе информации об аминокислотных положениях и прогностических переменных; и выбор подгруппы пептидных последовательностей, идентифицированных на основе набора прогнозов презентации для получения персонализированного средства для терапии злокачественной опухоли; где прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва по меньшей мере 0,1%, 0,1%-50% или не более 50%.
[0214] В рамках настоящего изобретения предусматривается способ, включающий обучение машинно-обучаемой модели для прогнозирования презентации пептидов HLA, где обучение включает ввод информации об аминокислотных положениях последовательностей HLA-пептидов, выделенных из одного или более комплексов HLA-пептид в клетке, экспрессирующей аллель HLA класса II, в модель для прогнозирования презентации HLA-пептид, с использованием компьютерного процессора; машинно-обучаемую модель для прогнозирования презентации пептидов HLA, содержащую: множество прогностических переменных, идентифицированных по меньшей мере на основе обучающих данных, которая включает: информацию о последовательностях пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; обучающую информацию о пептидных последовательностях, включающую информацию об аминокислотных положениях обучающих пептидов, где обучающая информация о пептидных последовательностях ассоциирована с белком HLA, экспрессируемым в клетках; и функцию, отражающую взаимосвязь между информацией об аминокислотных положениях, получаемую в качестве вводной информации, и вероятностью презентации, сгенерированную в качестве результата на основе информации об аминокислотных положениях и прогностических переменных.
[0215] В некоторых вариантах осуществления модель презентации имеет положительную прогностическую величину по меньшей мере 0,25 при частоте отзыва по меньшей мере 0,1%, 0,1%-50% или не более 50%.
[0216] В некоторых вариантах осуществления модель презентации имеет положительную прогностическую величину по меньшей мере 0,4 при частоте отзыва по меньшей мере 0,1%, 0,1%-50% или не более 50%.
[0217] В некоторых вариантах осуществления модель презентации имеет положительную прогностическую величину по меньшей мере 0,6 при частоте отзыва по меньшей мере 0,1%, 0,1%-50% или не более 50%.
[0218] В некоторых вариантах осуществления масс-спектрометрия представляет собой моноаллельную масс-спектрометрию.
[0219] В некоторых вариантах осуществления пептиды презентируются белком HLA, экспрессируемым в клетках, посредством аутофагии.
[0220] В некоторых вариантах осуществления пептиды презентируются белком HLA, экспрессируемым в клетках, посредством фагоцитоза.
[0221] В некоторых вариантах осуществления качество обучающих данных возрастает при использовании множества качественных показателей.
[0222] В некоторых вариантах осуществления множество параметров качества включает общее устранение контаминирующих пептидов, высокую оцененную интенсивность пика, высокую оценку и высокую точность массы.
[0223] В некоторых вариантах осуществления оцененная интенсивность пика составляет по меньшей мере 50%.
[0224] В некоторых вариантах осуществления оцененная интенсивность пика составляет по меньшей мере 60%.
[0225] В некоторых вариантах осуществления оценка составляет по меньшей мере 7.
[0226] В некоторых вариантах осуществления точность массы составляет не более 5 м.д.
[0227] В некоторых вариантах осуществления точность массы составляет не более 2 м.д.
[0228] В некоторых вариантах осуществления показатель расщепления основной цепи составляет по меньшей мере 5.
[0229] В некоторых вариантах осуществления показатель расщепления основной цепи составляет по меньшей мере 8.
[0230] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, экспрессируемым в клетках, представляют собой пептиды, презентируемые единичным иммунопреципитированным белком HLA, экспрессируемым в клетках.
[0231] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, экспрессируемым в клетках, представляют собой пептиды, презентируемые единичным экзогенным белком HLA, экспрессируемым в клетках.
[0232] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, экспрессируемым в клетках, представляют собой пептиды, презентируемые единичным рекомбинантным белком HLA, экспрессируемым в клетках.
[0233] В некоторых вариантах осуществления множество прогностических переменных включает прогностическую переменную аффинности пептид-HLA.
[0234] В некоторых вариантах осуществления множество прогностических переменных включает прогностическую переменную уровня экспрессии исходного белка.
[0235] В некоторых вариантах осуществления множество прогностических переменных включает прогностическую переменную способности пептида к расщеплению.
[0236] В некоторых вариантах осуществления обучающая информация о пептидных последовательностях включает последовательности пептидов, презентируемых белком HLA, которые включают пептиды, идентифицированные посредством поиска по отсутствию ферментной специфичности в базе данных пептидов без модификации. В некоторых вариантах осуществления пептиды, презентируемые белком HLA, включают пептиды, идентифицированные посредством поиска с использованием инструментов секвенирования белков de novo.
[0237] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, включают пептиды, идентифицированные посредством поиска в пептидной базе данных с использованием стратегии обратного поиска в базе данных.
[0238] В некоторых вариантах осуществления белок HLA включает белок HLA-DR и HLA-DP или HLA-DQ. В некоторых вариантах осуществления белок HLA включает белок HLA-DR, выбранный из группы, состоящей из белков HLA-DR, и HLA-DP или HLA-DQ. В некоторых вариантах осуществления белок HLA включает белок HLA-DR, выбранный из группы, состоящей из: HLA-DPB1*01:01/HLA-DPA1*01:03, HLA-DPB1*02:01/HLA-DPA1*01:03, HLA-DPB1*03:01/HLA-DPA1*01:03, HLA-DPB1*04:01/HLA-DPA1*01:03, HLA-DPB1*04:02/HLA-DPA1*01:03, HLA-DPB1*06:01/HLA-DPA1*01:03, HLA-DQB1*02:01/HLA-DQA1*05:01, HLA-DQB1*02:02/HLA-DQA1*02:01, HLA-DQB1*06:02/HLA-DQA1*01:02, HLA-DQB1*06:04/HLA-DQA1*01:02, HLA-DRB1*01:01, HLA-DRB1*01:02, HLA-DRB1*03:01, HLA-DRB1*03:02, HLA-DRB1*04:01, HLA-DRB1*04:02, HLA-DRB1*04:03, HLA-DRB1*04:04, HLA-DRB1*04:05, HLA-DRB1*04:07, HLA-DRB1*07:01, HLA-DRB1*08:01, HLA-DRB1*08:02, HLA-DRB1*08:03, HLA-DRB1*08:04, HLA-DRB1*09:01, HLA-DRB1*10:01, HLA-DRB1*11:01, HLA-DRB1*11:02, HLA-DRB1*11:04, HLA-DRB1*12:01, HLA-DRB1*12:02, HLA-DRB1*13:01, HLA-DRB1*13:02, HLA-DRB1*13:03, HLA-DRB1*14:01, HLA-DRB1*15:01, HLA-DRB1*15:02, HLA-DRB1*15:03, HLA-DRB1*16:01, HLA-DRB3*01:01, HLA-DRB3*02:02, HLA-DRB3*03:01, HLA-DRB4*01:01 и HLA-DRB5*01:01.
[0239] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, включают пептиды, идентифицированные посредством сравнения спектров MS/MS для HLA-пептидов со спектрами MS/MS для одного или более HLA-пептидов в пептидной базе данных.
[0240] В некоторых вариантах осуществления мутация выбрана из группы, состоящей из точковой мутации, мутации участка сплайсинга, мутации со сдвигом рамки считывания, мутации со сквозным прочитыванием и мутации со слиянием генов.
[0241] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, имеют длину 15-40 аминокислот.
[0242] В некоторых вариантах осуществления пептиды, презентируемые белком HLA, включают пептиды, идентифицированные посредством (a) выделения одного или более комплексов HLA из клеточной линии, экспрессирующей один аллель HLA класса II; (b) выделения одного или более HLA-пептидов из одного или более комплексов HLA; (c) получение спектров MS/MS для одного или более выделенных HLA-пептидов; и (d) получения пептидной последовательности, которая соответствует спектрам MS/MS одного или более выделенных HLA-пептидов, из базы данных пептидов; где одна или более последовательностей, полученных на стадии (d), идентифицирует последовательность одного или более выделенных HLA-пептидов.
[0243] В некоторых вариантах осуществления персонализированная терапия злокачественной опухоли дополнительно включает адъювант.
[0244] В некоторых вариантах осуществления персонализированная терапия злокачественной опухоли дополнительно включает ингибитор иммунной точки контроля.
[0245] В некоторых вариантах осуществления обучающие данные включают структурированные данные, данные временного ряда, неструктурированные данные, реляционные данные или любую их комбинацию.
[0246] В некоторых вариантах осуществления неструктурированные данные включают данные в виде изображений.
[0247] В некоторых вариантах осуществления реляционные данные включают данные системы потребителя, системы масштаба предприятия, операционной системы, веб-сайта, доступного через сеть прикладного программного интерфейса (API), или любой их комбинации.
[0248] В некоторых вариантах осуществления обучающие данные загружают в базу данных на основе облака.
[0249] В некоторых вариантах осуществления обучение проводят с использованием сверточных нейронных сетей.
[0250] В некоторых вариантах осуществления сверточные нейронные сети включают по меньшей мере два сверточных слоя.
[0251] В некоторых вариантах осуществления сверточные нейронные сети включают по меньшей мере одну стадию пакетной нормализации.
[0252] В некоторых вариантах осуществления сверточные нейронные сети включают по меньшей мере одну стадию пространственного исключения.
[0253] В некоторых вариантах осуществления сверточные нейронные сети включают по меньшей мере одну стадию глобального максимального пулинга.
[0254] В некоторых вариантах осуществления сверточные нейронные сети включают по меньшей мере один плотный слой.
[0255] В некоторых вариантах осуществления идентификация пептидных последовательности включает идентификацию пептидных последовательностей с мутацией, экспрессируемой в злокачественных клетках индивидуума.
[0256] В некоторых вариантах осуществления идентификация пептидных последовательностей включает идентификацию пептидных последовательностей, не экспрессируемых в нормальных клетках индивидуума.
[0257] В некоторых вариантах осуществления идентификация пептидных последовательностей включает идентификацию сверхэкспрессируемых пептидных последовательностей.
[0258] В некоторых вариантах осуществления идентификация пептидных последовательностей включает идентификацию вирусных пептидных последовательностей. В одном аспекте в рамках настоящего изобретения предусматривается способ идентификации пептидов, специфичных к HLA класса II, для иммунотерапии, специфической для индивидуума, причем способ включает: идентификацию пептида-кандидата, содержащего эпитоп; ввод информации об аминокислотах для множества пептидных последовательностей, каждая из которых содержит эпитоп, с использованием компьютерного процессора, в машинно-обучаемую модель для прогнозирования презентации пептидов HLA с получением набора прогнозов презентации посредством HLA пептидной последовательности иммунной клетке, причем каждый прогноз презентации отражает вероятность того, что один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, будет презентировать данную пептидную последовательность, содержащую эпитоп; где модель прогнозирования имеет положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва по меньшей мере 0,1%, 0,1%-50% или не более 50%, отбор белка из одного или более белков, кодируемых аллелем HLA класса II клетки индивидуума, спрогнозированных в качестве связывающихся с пептидом-кандидатом посредством модели прогнозирования, где белок имеет вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентировать пептид-кандидат иммунной клетке; приведение пептида-кандидата в контакт с белком, кодируемым аллелем HLA класса II, так чтобы пептид-кандидат конкурировал с пептидом-местозаполнителем, связанным с белком, кодируемым аллелем HLA класса II; и идентификацию пептида-кандидата в качестве пептида для иммунотерапии, специфичного к белку, кодируемому аллелем HLA класса II, на основе того, вытесняет ли пептид-кандидат пептид-местозаполнитель.
[0259] В некоторых вариантах осуществления иммунотерапия представляет собой иммунотерапию злокачественной опухоли.
[0260] В некоторых вариантах осуществления идентификация включает сравнение последовательностей ДНК, РНК или белков из злокачественных клеток индивидуума с последовательностями ДНК, РНК или белков из нормальных клеток индивидуума. В некоторых вариантах осуществления эпитоп представляет собой специфический для злокачественной опухоли эпитоп.
[0261] В некоторых вариантах осуществления по меньшей мере один белок, кодируемый аллелем HLA класса II, содержит по меньшей мере субъединицу альфа 1 и субъединицу бета 1 белка HLA или их фрагменты, присутствующие в форме димера. В некоторых вариантах осуществления пептид-местозаполнитель представляет собой пептид CLIP. В некоторых вариантах осуществления пептид-местозаполнитель представляет собой пептид CMV. В некоторых вариантах осуществления способ дополнительно включает измерение IC50 для вытеснения пептида-местозаполнителя пептидом-мишенью. В некоторых вариантах осуществления IC50 для вытеснения пептида-местозаполнитель пептидом-мишенью составляет менее 500 нМ. В некоторых вариантах осуществления по меньшей мере один белок из одного или более белков, кодируемых аллелем HLA класса II клетки индивидуума, представляет собой тетрамер или мультимер HLA класса II. В некоторых вариантах осуществления пептид-мишень далее идентифицируют посредством масс-спектрометрии. В некоторых вариантах осуществления по меньшей мере один белок, кодируемый аллелем HLA класса II клетки индивидуума, представляет собой рекомбинантный белок. В некоторых вариантах осуществления по меньшей мере один белок, кодируемый аллелем HLA класса II клетки индивидуума, экспрессируется в эукариотической клетке.
[0262] В одном аспекте в рамках настоящего изобретения предусматривается способ анализа для подтверждения специфичности пептида-кандидата в отношении связывания белка HLA класса II, причем способ включает: экспрессию в эукариотической клетке конструкции полинуклеиновой кислоты, содержащей последовательность нуклеиновой кислоты, кодирующую белок HLA класса II, содержащий альфа-цепь и бета-цепь или их части, способный связывать пептид, содержащий связывающий MHC-II эпитоп, и где экспрессированный белок HLA класса II или его часть остаются связанными с пептидом-местозаполнителем; выделение белка HLA класса II или его частей, экспрессируемых в эукариотической клетке; проведение анализа пептидного обмена посредством (a) добавления возрастающего количества пептида-кандидата для определения того, вытесняет ли пептид-кандидат пептид-местозаполнитель, ассоциированный с белком HLA класса II или его частями; и (b) вычисления IC50 реакции вытеснения для определения аффинности пептида-кандидата к белку HLA класса II или его частям относительно пептида-местозаполнителя, тем самым подтверждая специфичность пептида-кандидата в отношении связывания белка HLA класса II.
[0263] В некоторых вариантах осуществления идентичность пептида известна. В некоторых вариантах осуществления идентичность пептида неизвестна. В некоторых вариантах осуществления идентичность пептида определяют посредством масс-спектрометрии.
[0264] В некоторых вариантах осуществления анализ пептидного обмена включает детекцию пептидных флуоресцентных зондов или меток. В некоторых вариантах осуществления пептид-местозаполнитель представляет собой пептид CLIP.
[0265] В некоторых вариантах осуществления конструкция полинуклеиновой кислоты содержит экспрессирующий вектор, дополнительно содержащий одно или более из: промотора, линкера, одного или более участков расщепления протеазой, секреторного сигнала, факторов димеризации, последовательности рибосомального пропускания, одной или более меток для очистки и/или детекции.
[0266] В одном аспекте в рамках настоящего изобретения предусматривается способ анализа иммуногенности пептида, связывающего MHC класса II, причем способ включает: выбор белка, кодируемого аллелем HLA класса II, спрогнозированного посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA в качестве связывающего пептид; где модель прогнозирования имеет положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва по меньшей мере 0,1%, 0,1%-50% или не более 50%, и где белок имеет вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентировать идентифицированную пептидную последовательность; приведение пептида в контакт с выбранным белком, кодируемым аллелем HLA класса II, так чтобы пептид конкурировал с пептидом-местозаполнителем, ассоциированным с выбранным белком, кодируемым аллелем HLA класса II, и вытеснял пептид-местозаполнитель, тем самым формируя комплекс, содержащий белок HLA класса II и идентифицированный пептид; приведение белка HLA класса II и идентифицированного пептидного комплекса в контакт с CD4+ T-клеткой, анализ одного или более параметров активации CD4+T-клетки, выбранных из индукции цитокина, индукции хемокина и экспрессии маркера клеточной поверхности.
[0267] В некоторых вариантах осуществления аллель HLA класса II представляет собой тетрамер или мультимер. В некоторых вариантах осуществления цитокин представляет собой IL-2. В некоторых вариантах осуществления цитокин представляет собой IFN-гамма.
[0268] В одном аспекте в рамках настоящего изобретения предусматривается способ индукции активации CD4+ T-клеток у индивидуума для иммунотерапии злокачественной опухоли, причем способ включает: идентификацию пептидной последовательности, ассоциированной со злокачественной опухолью, содержащей мутацию злокачественной опухоли, где идентификация включает сравнение последовательностей ДНК, РНК или белков из злокачественных клеток индивидуума с последовательностями ДНК, РНК или белков из нормальных клеток индивидуума; выбор белка, кодируемого аллелем HLA класса II, который в норме экспрессируется клеткой индивидуума и спрогнозирован посредством машинно-обучаемой модели для прогнозирования презентации пептидов HLA в качестве связывающегося с пептидом; где модель прогнозирования имеет положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва по меньшей мере 0,1%, 0,1%-50% или не более 50% и где белок имеет вероятность, превышающую пороговую величину вероятности прогнозирования презентации, презентировать идентифицированную пептидную последовательность; приведение идентифицированного пептида в контакт с отобранным белком, кодируемым аллелем HLA класса II, для подтверждения того, конкурирует ли идентифицированный пептид с пептидом-местозаполнителем, ассоциированным с отобранным белком, кодируемым аллелем HLA класса II, для вытеснения пептида-местозаполнителя с величиной IC50 менее 500 нМ; очистку идентифицированного пептида; и введение эффективного количества идентифицированного пептида индивидууму.
[0269] В одном аспекте в рамках настоящего изобретения предусматривается способ производства тетрамеров или мультимеров HLA класса II, причем способ включает: экспрессию в эукариотической клетке вектора, содержащего последовательность нуклеиновой кислоты, кодирующую альфа-цепь и бета-цепь белка HLA, линкер, один или более участков расщепления протеазой, секреторный сигнал, мотив биотинилирования и по меньшей мере одну метку для идентификации или очистки, так чтобы каждый гетеродимер альфа 1 и бета 1 белка HLA секретировался в димеризованном состоянии, где гетеродимер связан с пептидом-местозаполнителем, очистку секретируемого гетеродимера из клеточной среды, подтверждение активности связывания пептида с использованием анализа пептидного обмена, добавление стрептавидина, тем самым осуществляя конъюгацию гетеродимеров тетрамеры, очистку тетрамеров и получение выхода более 1 мг/л.
[0270] В некоторых вариантах осуществления вектор содержит промотор CMV. В некоторых вариантах осуществления вектор содержит последовательность, кодирующую пептид-местозаполнитель, связанный через расщепляемый участок с цепью бета 1. В некоторых вариантах осуществления анализ пептидного обмена вовлекает предшествующее отщепление пептида-местозаполнителя от бета-цепи. В некоторых вариантах осуществления расщепляемый участок представляет собой участок расщепления тромбином. В некоторых вариантах осуществления анализ пептидного обмена представляет собой анализ FRET. В некоторых вариантах осуществления очистку проводят посредством любой из: колоночной хроматографии, периодической хроматографии, ионообменной хроматографии, эксклюзионной хроматографии, аффинной хроматографии или LC-MS.
[0271] В одном аспекте в рамках настоящего изобретения предусматривается композиция, содержащая тетрамеры HLA класса II, содержащие гетеродимеры либо HLA-DR, либо HLA-DP, либо HLA-DQ, причем каждый гетеродимер содержит альфа- и бета-цепь, очищенные и присутствующие в концентрации более 0,25 мг/л. В некоторых вариантах осуществления тетрамер HLA касса II содержит пары гетеродимеров, выбранные из группы, состоящей из следующих: белок может быть выбран из группы, состоящей из белка HLA-DR, и HLA-DP или HLA-DQ. В некоторых вариантах осуществления белок HLA выбран из группы, состоящей из: HLA-DPB1*01:01/HLA-DPA1*01:03, HLA-DPB1*02:01/HLA-DPA1*01:03, HLA-DPB1*03:01/HLA-DPA1*01:03, HLA-DPB1*04:01/HLA-DPA1*01:03, HLA-DPB1*04:02/HLA-DPA1*01:03, HLA-DPB1*06:01/HLA-DPA1*01:03,HLA-DQB1*02:01/HLA-DQA1*05:01,HLA-DQB1*02:02/HLA-DQA1*02:01, HLA-DQB1*06:02/HLA-DQA1*01:02,HLA-DQB1*06:04/HLA-DQA1*01:02, HLA-DRB1*01:01, HLA-DRB1*01:02, HLA-DRB1*03:01, HLA-DRB1*03:02, HLA-DRB1*04:01, HLA-DRB1*04:02, HLA-DRB1*04:03, HLA-DRB1*04:04, HLA-DRB1*04:05, HLA-DRB1*04:07, HLA-DRB1*07:01, HLA-DRB1*08:01, HLA-DRB1*08:02, HLA-DRB1*08:03, HLA-DRB1*08:04, HLA-DRB1*09:01, HLA-DRB1*10:01, HLA-DRB1*11:01, HLA-DRB1*11:02, HLA-DRB1*11:04, HLA-DRB1*12:01, HLA-DRB1*12:02, HLA-DRB1*13:01, HLA-DRB1*13:02, HLA-DRB1*13:03, HLA-DRB1*14:01, HLA-DRB1*15:01, HLA-DRB1*15:02, HLA-DRB1*15:03, HLA-DRB1*16:01, HLA-DRB3*01:01, HLA-DRB3*02:02, HLA-DRB3*03:01, HLA-DRB4*01:01, HLA-DRB5*01:01).
[0272] В некоторых вариантах осуществления пары гетеродимеров экспрессируются в эукариотической клетке. В некоторых вариантах осуществления пара гетеродимеров кодируется вектором. В некоторых вариантах осуществления вектор содержит: последовательность нуклеиновой кислоты, кодирующую альфа-цепь и бета-цепь белка HLA, секреторный сигнал, мотив биотинилирования и по меньшей мере одну метку для идентификации или для очистки, так что каждый гетеродимер альфа 1 и бета 1 белка HLA секретируется в димеризованном состоянии, где секретируемый гетеродимер ассоциирован с пептидом-местозаполнителем. В некоторых вариантах осуществления вектор содержит: последовательность нуклеиновой кислоты, кодирующую альфа-цепь и бета-цепь белка HLA, секреторный сигнал, мотив биотинилирования и по меньшей мере одну метку для идентификации или для очистки, так что каждый гетеродимер альфа 1 и бета 1 белка HLA секретируется в димеризованном состоянии, где секретируемый гетеродимер ассоциирован с пептидом-местозаполнителем.
[0273] В некоторых вариантах осуществления гетеродимеры HLA класса II секретируются из эукариотических клеток в среду для культивирования клеток, и их очищают посредством любой из: колоночной или периодической хроматографии, ионообменной хроматографии, эксклюзионной хроматографии, аффинной хроматографии или LC-MS.
[0274] В одном аспекте в рамках настоящего изобретения предусматривается способ скрининга лекарственного средства, содержащего полипептидную последовательность, в отношении иммуногенности у индивидуума, причем способ включает: ввод информации об аминокислотах для пептидных последовательностей полипептидной последовательности с использованием компьютерного процессора в машинно-обучаемую модель для прогнозирования презентации пептидов HLA с получением набора прогнозов презентации для пептидных последовательностей, причем каждый прогноз презентации отражает вероятность того, что один или более белков, кодируемых аллелем HLA класса I или II в клетке индивидуума будет презентировать последовательность эпитопа данной пептидной последовательности; где машинно-обучаемая модель для прогнозирования презентации пептидов HLA включает: множество прогностических переменных, идентифицированных по меньшей мере на основе обучающих данных где обучающие данные включают: информацию о последовательностях пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; обучающую информацию о пептидных последовательностях, включающую информацию об аминокислотных положениях, где обучающая информация о пептидных последовательностях ассоциирована с белком HLA, экспрессируемым в клетках; и функцию, отражающую взаимосвязь между информацией об аминокислотных положениях, полученную в качестве вводной информации, и вероятностью презентации, сгенерированную в качестве результата на основе информации об аминокислотных положениях и прогностических переменных; (b) определение или прогнозирование того, что каждая из пептидных последовательностей полипептидной последовательности не будет иммуногенной для индивидуума на основе набора прогнозов презентации; и (c) введение индивидууму композиции, содержащей лекарственное средство.
[0275] В одном аспекте в рамках настоящего изобретения предусматривается способ скрининга лекарственного средства, содержащего полипептидную последовательность, в отношении иммуногенности у индивидуума, причем способ включает: (a) ввод информации об аминокислотах для пептидных последовательностей полипептидной последовательности с использованием компьютерного процессора в машинно-обучаемую модель для прогнозирования презентации пептидов HLA с получением набора прогнозов презентации для пептидных последовательностей, причем каждый прогноз презентации отражает вероятность того, что один или более белков, кодируемых аллелем HLA класса I или II клетки индивидуума, будет презентировать эпитопную последовательность данной пептидной последовательности; где машинно-обучаемая модель для прогнозирования презентации пептидов HLA включает: множество прогностических переменных, идентифицированных по меньшей мере на основе обучающих данных; где обучающие данные включают: последовательность информацию о последовательности пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; обучающую информацию о пептидных последовательностях, включающую информацию об аминокислотных положениях, где обучающая информация о пептидных последовательностях ассоциирована с белком HLA, экспрессируемым в клетках; и функцию, отражающую взаимосвязь между информацией об аминокислотных положениях, полученной в качестве вводной информации, и вероятностью презентации, сгенерированную в качестве результата на основе информации об аминокислотных положениях и прогностических переменных; (b) определение или прогнозирование того, что по меньшей мере одна из пептидных последовательностей полипептидной последовательности будет иммуногенной на основе набора прогнозов презентации.
[0276] В одном варианте осуществления способ дополнительно включает решение не вводить лекарственное средство индивидууму.
[0277] В одном варианте осуществления лекарственное средство включает антитело или его антигенсвязывающий фрагмент.
[0278] В одном варианте осуществления каждая из пептидных последовательностей полипептидных последовательностей содержит непрерывную пептидную последовательность полипептидной последовательности, которая имеет длину 8, 9, 10, 11 или 12 аминокислот, и где белок, кодируемый аллелем HLA класса I или II клетки индивидуума, представляет собой белок, кодируемый аллелем HLA класса I индивидуума.
[0279] В одном варианте осуществления каждая из пептидных последовательностей полипептидных последовательностей содержит непрерывную пептидную последовательность полипептидной последовательности, которая имеет длину 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25 аминокислот, и где белок, кодируемый аллелем HLA класса I или II клетки индивидуума, кодируется аллелем MHC класса II клетки индивидуума.
[0280] В одном аспекте в рамках настоящего изобретения предусматривается способ лечения индивидуума с аутоиммунным заболеванием или состоянием, включающий: (a) идентификацию или прогнозирование эпитопа экспрессируемого белка, презентируемого HLA класса I или II клетки индивидуума, где на комплекс, содержащий идентифицированный или спрогнозированный эпитоп и HLA класса I или II, нацеливается CD8 или CD4 T-клетка индивидуума; (b) идентификацию T-клеточного рецептора (TCR), который связывается с комплексом; (c) экспрессию TCR в регуляторной T-клетке от индивидуума или аллогенной регуляторной T-клетке; и (d) введение регуляторной T-клетки, экспрессирующей TCR, индивидууму.
[0281] В одном варианте осуществления аутоиммунное заболевание или состояние представляет собой диабет.
[0282] В одном варианте осуществления клетка представляет собой островковую клетку.
[0283] В одном аспекте в рамках настоящего изобретения предусматривается способ лечения индивидуума с аутоиммунным заболеванием или состоянием, включающий введение индивидууму регуляторной T-клетки, экспрессирующей T-клеточный рецептор (TCR), который связывается с комплексом, содержащим (i) эпитоп экспрессируемого белка, идентифицированный или спрогнозированный в качестве презентируемого посредством HLA класса I или II клетки индивидуума, и (ii) HLA класса I или II, где на комплекс нацелена CD8 или CD4 T-клетка индивидуума.
[0284] Дополнительные аспекты и преимущества настоящего изобретения станут хорошо понятными специалистам в данной области из приведенного ниже подробного описания, где только иллюстративные варианты осуществления настоящего изобретения представлены и описаны. Как будет понятно, могут существовать другие и отличающиеся варианты осуществления настоящего изобретения, и некоторые из его деталей могут быть модифицированы в различным очевидным образом, во всех случаях без отклонения от изобретения. Таким образом, чертежи и описание следует считать иллюстративными по своей природе, а не ограничивающими.
[0285] MAPTAC™ может использоваться для высокопроизводительного анализа связывания пептидов, где пептиды, связанные с HLA класса II, количественно определяют после выделения с использованием конструкций MAPTAC™ в различные моменты времени и в различных условиях, таких как нагревание при 37°C, для получения последовательностей популяций пептидов с различной стабильностью с использованием LC-MS/MS.
[0286] В одном аспекте в рамках настоящего изобретения предусматривается способ лечения злокачественной опухоли у индивидуума, причем способ включает: идентификацию пептидных последовательностей, где пептидные последовательности ассоциированы со злокачественной опухолью, где идентификация включает сравнение последовательностей ДНК, РНК или белков из злокачественных клеток индивидуума с последовательностями ДНК, РНК или белков из нормальных клеток индивидуума; ввод информации об аминокислотах для идентифицированных пептидных последовательностей с использованием компьютерного процессора в машинно-обучаемую модель для прогнозирования презентации пептидов HLA с получением набора прогнозов презентации для идентифицированных пептидных последовательностей, причем каждый прогноз презентации отражает вероятность того, что один или более белков, кодируемых аллелем HLA класса II индивидуума, будет презентировать данную последовательность идентифицированной пептидной последовательности; где машинно-обучаемая модель для прогнозирования презентации пептидов HLA включает: множество прогностических переменных, идентифицированных по меньшей мере на основе обучающих данных где обучающие данные включают: информацию о последовательности пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; обучающую информацию о пептидных последовательностях, включающую информацию об аминокислотных положениях, где обучающая информация о пептидных последовательностях ассоциирована с белком HLA, экспрессируемым в клетках; и функцию, отражающую взаимосвязь между информацией об аминокислотных положениях, полученную в качестве вводной информации, и вероятностью презентации, сгенерированную в качестве результата на основе информации об аминокислотных положениях и прогностических переменных; и выбор подгруппы пептидных последовательностей, идентифицированных на основе набора прогнозов презентации для получения персонализированного средства терапии злокачественной опухоли; и введение индивидууму композиции, содержащей один или более пептидов, где прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва по меньшей мере 0,1%, 0,1%-50% или не более 50%.
[0287] В некоторых вариантах осуществления машинно-обучаемая модель для прогнозирования презентации пептидов HLA включает информацию о последовательности для последовательностей пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии, после проведения обращенно-фазового фракционирования вне системы.
[0288] В некоторых вариантах осуществления прогностическая модель демонстрирует улучшение в от 1,1× до 100× по сравнению с NetMHCIIpan. В некоторых вариантах осуществления прогностическая модель демонстрирует улучшение в 1,1, 2, 3, 4, 5, 6, 7, 7,4, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 18, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 50, 55, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 8, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99, 100 раз по сравнению с NetMHCIIpan.
ВКЛЮЧЕНИЕ В КАЧЕСТВЕ ССЫЛКИ
[0289] Все публикации, патенты и патентные заявки, упоминаемые в настоящем описании, включены в настоящее описание в качестве ссылок в той же степени, как если бы было конкретно и индивидуально указано, что каждая индивидуальная публикация, патент или патентная заявка включены в качестве ссылок. Если публикации и патенты или патентные заявки, включенные в качестве ссылок, противоречат описанию, предусматривается, что описание преобладает и/или имеет приоритет над таким противоречащим материалом.
КРАТКОЕ ОПИСАНИЕ ЧЕРЖЕТЕЙ
[0290] Новые признаки изобретения указаны детально в прилагаемой формуле изобретения. Лучшее понимание признаков и преимуществ настоящего изобретения будет достигнуто с помощью следующего подробного описания, в которому казаны иллюстративные варианты осуществления, в которых используются принципы изобретения, и прилагаемых чертежей (также "фиг." в настоящем описании), на которых:
[0291] На фиг. 1A представлена диаграмма, демонстрирующая пептид, состыкованный с белком MHC класса I. На фиг. представлена SEQ ID NO: 36.
[0292] На фиг. 1B представлена иллюстративная диаграмма, демонстрирующая пептид, состыкованный с белком MHC класса II. На фиг. представлена SEQ ID NO: 37.
[0293] На фиг. 2 представлен иллюстративный экспериментальный подход для получения данных о связывании пептидов для моноаллельного HLA класса II. Пептиды HLA класса II вводят в клетку, включая клетку, не экспрессирующую HLA класса II, так чтобы определенный аллель(и) HLA класса II экспрессировался в клетке. Популяции генно-модифицированных экспрессирующих HLA клеток собирают, лизируют и их комплексы HLA-пептид метят (например, биотинилируют) и подвергают иммунной очистке (например, с использованием взаимодействия биотин-стрептавидин). Ассоциированные с HLA пептиды, специфичные к единичному HLA, можно элюировать из их меченых (например, биотинилированных) комплексов и оценивать (например, секвенировать с использованием LC-MS/MS высокого разрешения).
[0294] На фиг. 3 представлено иллюстративное логотипное представление последовательности пептидов, связанных с HLA класса II-DRB1*11:01, для следующих случаев: клеточная линия Neon BAP, Expi293; клеточная линия Neon BAP, A375; IEDB, аффинность <50 нМ; и общие Ab против HLA класса II, гомозиготные LCL. На фиг. 3 показано, что примеры полученных посредством MS мотивов соответствуют известным паттернам и демонстрируют неизменность среди трансфицированных клеточных линий.
[0295] На фиг. 4 представлено иллюстративное изображение эффективности прогнозирования связывания HLA класса II. На фиг. 4 представлена столбиковая диаграмма, демонстрирующая эффективность средства прогнозирования связывания (neonmhc2) и NetMHCIIpan, применяемых для валидирующего набора данных, состоящего из наблюдаемых посредством масс-спектрометрии пептидов и пептидов-ловушек, которые получены в соотношении 1:19 (наилучшие последовательности:ловушки) посредством случайного шаффлинга наиболее подходящих пептидов. Для средства прогнозирования NEON neonmhc2 сконструирована отдельная модель для каждого показанного аллеля MHC II. Высота столбиков показывает положительную прогностическую величину (PPV), определяемую как долю срогнозированных связывающих молекул в валидирующем наборе, которые в действительности были наиболее подходящими пептидами. Аллели сортировали в соответствии с эффективностью модели при прогнозировании для этого аллеля.
[0296] На фиг. 5 представлен иллюстративный эффект пороговых значений оцененной интенсивности пика (SPI) на валидацию средства прогнозирования связывания. На фиг. 5 представлена эффективность средства прогнозирования связывания HLA класса II после обучения/валидации на наборах пептидов с различными предельными значениями оцененной интенсивности пика (SPI). Для каждой обученной аллелеспецифической модели представлена эффективность модели в 3 условиях: обучение и оценка на наборах данных с использованием выявленных посредством MS наиболее подходящих пептидов с SPI более чем или ровно 70, обучение на пептидах SPI с более чем или ровно 50 и валидация на пептидах с SPI более чем или ровно 70, и обучение и валидация на пептидах с SPI более чем или ровно 50.
[0297] На фиг. 6 представлена иллюстративная столбиковая диаграмма, демонстрирующая репрезентативные данные для ряда выявленных пептидов посредством определения аллельного профиля с использованием LC-MS/MS с предельными значениями оцененной интенсивности пика (SPI) более или ровно 70. Каждый столбик соответствует общему количеству выявленных пептидов для аллеля. Представлены собранные данные для 35 аллелей HLA-DR. Собранные данные для 35 аллелей HLA-DR имеют популяционный охват >95% для HLA-DR (частоты аллелей в США).
[0298] На фиг. 7A представлена PPV модели при применении для тестовой части данных для указанных аллелей HLA класса II. Использованные пептиды-ловушки представляли собой перемешанные последовательности положительных (наиболее подходящих) пептидных последовательностей в соотношении наиболее подходящих последовательностей и ловушек 1:19. PPV была определена посредством идентификации 5% пептидов с наилучшей оценкой в тестовой части и определения их доли, которые были положительными в отношении связывания с белком, кодируемым соответствующем аллелем HLA класса II.
[0299] На фиг. 7B представлена иллюстративная эффективность прогнозирования в качестве функции размера обучающего набора (кривые, полученные посредством искусственного уменьшения выборки для обучающего набора). На фиг. 7B показано, что, главным образом, для 35 полученных аллелей HLA-DR, когда размер обучающей выборки возрастает, величина PPV возрастает.
[0300] На фиг. 8 представлен иллюстративный график, демонстрирующий, что связанные с процессингом переменные могут далее улучшить прогнозирование. Могут быть различены случайные последовательности выявленных посредством MS пептидов из кодирующего белки экзома. На обучающей части данных логистическая регрессия может быть аппроксимирована для прогнозирования презентации HLA класса II с использованием силы связывания (средство прогнозирования NetMHCIIpan или Neon) и показателей процессинга (экспрессия RNA-Seq и установленная составляющая смещения для генного уровня). На отдельной оценочной части положения экзонов, перекрывающие выявленные посредством MS пептиды MHC II ("наиболее подходящие последовательности") могут быть оценены вместе со случайными экзонными положениями, не выявленными при MS (соотношение 1:499). Наивысшие 0,2% (1/500) могут быть названы положительными результатами, и из этого порогового значения может оценена положительная прогностическая величина.
[0301] На фиг. 9 представлена иллюстративная архитектура нейронной сети. Вводимые пептиды представлены в качестве 20-меров, причем более короткие пептиды дополнены символами "отсутствия". Каждый пептид имеет 31-мерное вложение, так что входные данные для нейронной сети представляют собой матрицу 20×31. Перед обработкой нейронной сетью проводится нормализация признаков на матрице 20×31 на основе средних значений величин признаков и стандартных отклонений в обучающем наборе. Первый сверточный слой имеет ядро из 9 аминокислот и 50 фильтров (также называемых каналами) с активационной функцией блока линейной ректификации (ReLU). После этого следует пакетная нормализация, затем пространственное исключение с частотой исключения 20%. После этого следует другой сверточный слой с ядром из 3 аминокислот и 20 фильтрами с функцией активации ReLU, за которыми вновь следует пакетная нормализация и пространственное исключение с частотой исключения 20%. Затем применяется глобальный максимальный пулинг, учитывая максимально активированный нейрон в каждом из 20 фильтров; затем эти 20 величин передаются в полносвязанный (плотный) слой с единичным нейроном с использованием сигмовидной функции активации. Результат этого слоя обрабатывается в качестве прогноза связывание/не связывание. Регуляризацию L2 применяют для веса первого сверточного слоя, второго сверточного слоя и плотного слоя с весом 0,05, 0,1 и 0,01, соответственно. Дополнительные используемые модели варьируют количество сверточных слоев и размер ядра для каждого слоя.
[0302] На фиг. 10 представлена иллюстративная компьютерная система контроля, которая запрограммирована или иным образом организована для осуществления способов, описанных в настоящем описании.
[0303] На фиг. 11A представлен иллюстративный обзор экспериментального процесса MAPTAC™. На фиг. представлена SEQ ID NO: 38.
[0304] На фиг. 11B представлены иллюстративные количества пептидов на аллель, объединенные для повторений.
[0305] На фиг. 11C представлены иллюстративные распределения длин пептидов для аллелей HLA класса I и HLA класса II, профили которых были определены посредством MAPTAC™.
[0306] На фиг. 11D представлены иллюстративные частоты остатков цистина, наблюдаемые посредством MAPTAC™ и IEDB (аллели DRB1*01:01, DRB1*03:01, DRB1*09:01, и DRB1*11:01), протеома человека и данных мульталлельной MS из предшествующих публикаций.
[0307] На фиг. 12A представлены частоты у европиоидов аллелей HLA-DR, -DP и -DQ, присутствующих у >1% индивидуумов, и количества пептидов из указанных источников, определенных в качестве пептидов с сильным связыванием (<50 нМ).
[0308] На фиг. 12B представлено иллюстративное распределение длин пептидов IEDB с ассоциированными показателями аффинности в отношении HLA класса II.
[0309] На фиг. 12C представлены иллюстративные вестерн-блоты для клеточных линий (1) Expi293, (2) HeLa и (3) A375, индивидуально трансфицированных двумя аллелями HLA класса I и двумя аллелями HLA класса II: HLA-A*02:01, HLA-B*45:01, HLA-DRB1*01:01 и HLA-DRB1*11:01. Мембраны подвергали блоттингу с антителом против эпитопной метки с биотинлигазой для визуализации биотин-акцепторного пептида (BAP) и антителом против бета-тубулина в качестве контроля загрузки. Дорожки соответствуют следующим фракциям, полученным в ходе протокола MAPTAC™: дорожка 1 исходный материал, дорожка 2 биотинилированный исходный материал, и дорожка 3 исходный материал после соосаждения.
[0310] На фиг. 12D представлены иллюстративные частоты аминокислот на остаток, наблюдаемые посредством MAPTAC™ и IEDB (аллели DRB1*01:01, DRB1*03:01, DRB1*09:01, и DRB1*11:01), протеома человека, и данных мультиаллельной MS из предшествующих публикаций.
[0311] На фиг. 12E представлены частоты для европиоидов аллелей HLA-DR, -DP и -DQ, присутствующих у >1% индивидуумов и количества пептидов из указанных источников, определенных в качестве молекул с сильным связыванием (<50 нМ). Эта фигура включает дополнительные данные относительно фиг. 12A. Дополнительные данные были получены с: tools.iedb.org/main/datasets/.
[0312] На фиг. 12F представлены иллюстративные частоты аминокислот на остаток, выявленные посредством MAPTAC™ (восстановленные и алкилированные), MAPTAC™ (без обработки) и IEDB (аллели DRB1*01:01, DRB1*03:01, DRB1*09:01 и DRB1*11:01), протеома человека и данных мультиаллельной MS из предшествующих публикаций.
[0313] На фиг. 13 изображено иллюстративное представление логотипов центральных связывающих последовательностей для аллелей MHC II согласно MAPTAC™ и IEDB. Логотипы последовательностей представляют собой графические представления, где высота каждой аминокислоты пропорциональна ее частоте встречаемости в пептиде, который связывается с белком MHC, кодируемым аллелем. Положения с наименьшей энтропией указаны цветом, где цвета соответствуют свойствам аминокислот. Пептиды происходят из указанных наборов данных и выровнены в соответствии со средством прогнозирования на основе CNN (способы). Логотипы соответствуют всем пептидам, включая пептиды, которые не в высокой степени соответствуют общему мотиву (например, пептиды не помещаются в кластер "мусора").
[0314] На фиг. 14A представлены иллюстративные логотипы последовательностей для связывающих HLA-A*02:01 пептидов (лиганды), проанализированных с использованием различных технологий определения профилей HLA-лиганд, включая анализы связывания, анализы стабильности, масс-спектрометрию растворимого HLA (sHLA), моноаллельную масс-спектрометрию и MAPTAC™ в двух различных клеточных линиях (A375 и expi293).
[0315] На фиг. 14B представлена иллюстративная доля пептидов MAPTAC™, демонстрирующих 0, 1, 2, 3 и 4 эвристически определенных якорей.
[0316] На фиг. 14C представлено иллюстративное распределение спрогнозированной посредством NetMHCIIpan аффинности связывания для выявленных посредством MAPTAC™ пептидов (20 пептидов на аллель, каждый с SPI>70 и размером вложенного множества >=2) и соответствующих по длине ловушек, взятых из протеома.
[0317] На фиг. 15A представлена иллюстративная архитектура сверточной нейронной сети (CNN), обученной для отличения моноаллельных пептидов MHC от перемешанных соответствующих по длине ловушек. На схеме указано использование вложения характеристик аминокислот, 2 сверточных слоев с различными размерами фильтров, и использование глобального максимального пулинга в качестве ввода для конечного логистического узла вывода.
[0318] На фиг. 15B представлен иллюстративный результат, который демонстрирует тау-статистику Кендалла для корреляции измеренной аффинности в IEDB с прогнозами связывания либо из neonmhc2, либо из NetMHCIIpan. Оцененные пептиды включают только те, которые были внесены в IEDB через год после выпуска NetMHCIIpan.
[0319] На фиг. 16 представлено иллюстративное изображение эффективности neonmhc2 в качестве функции размера обучающего набора данных.
[0320] На фиг. 17A представлено иллюстративное распределение на кластеры для MAPTAC™ (20 на аллель) внесенных в наборы данных MS, общие для DR и общие для MHC класса II. Наборы данных подвергали обратной свертке с использованием GibbsCluster. Каждая рамка соответствует одному пептиду MAPTAC™. Цвет рамки указывает на то, к какому кластеру он был отнесен, и серые столбики указывают на то, из какого аллеля пептид фактически произошел. Общее количество кластеров в кластерном решении Gibbs (правая сторона) было выбрано с использованием параметра взаимной информации (MI). Показатель MI также определяет, как образцы сортируют; образцы с решениями с высокими показателями MI представлены сверху.
[0321] На фиг. 17B представлены иллюстративные логотипы центральных связывающих последовательностей для данных мультиаллельной MS, подвергнутых обратной свертке посредством GibbsCluster. Каждый набор пептидов соответствует кластеру, который наилучшим образом выравнивался с внесенными из MAPTAC™ пептидами.
[0322] На фиг. 17C представлена репрезентативная эффективность моделей с использованием либо данных MAPTAC™, либо подвергнутых обратной свертке мультиаллельных данных для прогнозирования контрольных пептидов MAPTAC™. Для каждого аллеля больший из двух источников данных (обычно MAPTAC™) подвергали уменьшению выборки, так чтобы средства прогноза были основаны на равном количестве обучающих примеров. В качестве дополнительного сравнения представлена эффективность NetMHCIIpan.
[0323] На фиг. 17D представлены иллюстративные логотипы центральных связывающих последовательностей, происходящих из данных мультиаллельной MS из указанных источников.
[0324] На фиг. 18A представлен иллюстративный график доли пептидов против источника экспрессии генов (количество транскриптов на миллион (TPM)) для выявленных посредством MS пептидов и случайных протеомных ловушек (данные, повторно нанесенные на график из Schuster et al., 2017).
[0325] На фиг. 18B представлено иллюстративное выявленное против ожидаемого количество пептидов класса II на ген при определении посредством объединенного анализа наборов данных для рака ободочной и прямой кишки, меланомы и рака яичника (Löffler et al., 2018, и Schuster et al., 2017). Ожидаемое количество получено путем умножения длины гена на уровень экспрессии. Ожидаемые и выявленные количества суммировали для соответствующих выборок. Гены с известным присутствием в плазме указаны в соответствии с их концентрацией (вкладка).
[0326] На фиг. 18C представлено иллюстративное распределение показателей увеличения представленности (соотношение наблюдаемых и ожидаемых наблюдений, как на фиг. 18B) для генов, ассоциированных с аутофагией.
[0327] На фиг. 18D представлено иллюстративное распределение показателей увеличения представленности в зависимости от локализации каждого гена-источника. Локализацию гена-источника определяли с использованием Uniprot (uniprot_sprot.dat).
[0328] На фиг. 18E представлены иллюстративные данные, соответствующие сравнению ожидаемой против наблюдаемой частоты для доли от общего количества пептидов, обладающих аффинностью связывания MHC-II, сегрегированных на основе их свойств клеточной локализации.
[0329] На фиг. 18F представлены иллюстративные репрезентативные данные относительного соответствия пептидов в наблюдениях в отношении двух различных профилей экспрессии генов. Для каждого образца количества пептидов на генном уровне моделировали в качестве линейной комбинации совокупной опухолевой экспрессии генов и профиля экспрессии генов профессиональными APC (макрофаги). Соотношение коэффициентов определяет относительное соответствие каждого профиля экспрессии репертуару пептидов. Планки погрешности соответствуют 95% доверительному интервалу, вычисленному посредством бутстрэп-перевыборки.
[0330] На фиг. 19A представлены иллюстративные репрезентативные данные уровней экспрессии HLA-DRB1 в пяти иллюстративных испытаниях. Каждая точка соответствует экспрессии в индивидуальном типе клеток у индивидуального пациента, усредненной для клеток.
[0331] На фиг. 19B представлены иллюстративные репрезентативные данные экспрессии HLA-DRB1 в опухоли и строме в качестве входных данных, полученных посредством RNA-Seq для пациентов TCGA. Горизонтальные планки соответствуют индивидуальным пациентам, и они сгруппированы по типу опухоли. Пациентов включали, если они имели мутацию в гене каскада HLA класса II (CIITA, CD74 или CTSSS) при определении по сигналам мутаций на основе ДНК. Для каждого пациента долю экспрессии HLA-DRB1, отнесенной к опухолевой, оценивали в качестве min(1,2f), где f представляет собой долю чтений RNA-Seq в CIITA, CD74 или CTSS, демонстрирующих мутацию.
[0332] На фиг. 19C представлены иллюстративные репрезентативные данные дополнительных исследований RNA-Seq для единичных клеток, которые включают биоптаты до и после иммунотерапии с блокадой точки контроля.
[0333] На фиг. 20 представлены иллюстративные репрезентативные экспериментальные данные, оценивающие прогнозирование общей эффективности на натуральных донорских тканях.
[0334] На фиг. 21A представлены иллюстративные репрезентативные данные, демонстрирующие, что интегрированная модель презентации прогнозирует клеточные лигандомы HLA класса II. Они отображают PPV при соотношении наиболее подходящей последовательности и ловушки 1:499 для общих наборов данных для DR (также проанализированных на фиг. 30B и фиг. 32E). Средства прогноза используют прогнозирование связывания (NetMHCIIpan или neonmhc2) и необязательно используют экспрессию генов, смещение генов (согласно фиг. 32A) и перекрывание с ранее наблюдаемыми пептидами HLA-DQ. Для каждого пептида-кандидата показатель связывания вычисляли в качестве максимума среди аллелей HLA-DR в генотипе образца.
[0335] На фиг. 21B представлены иллюстративные репрезентативные данные, демонстрирующие эффективность прогнозирования для происходящих из опухоли пептидов, как идентифицировано с использованием SILAC, презентируемых дендритными клетками (проанализированными из клеточных лизатов) с использованием того же соотношения наиболее подходящая последовательность: ловушка и параметров эффективности, что и на фиг. 21A, с использованием или без использования показателей процессинга.
[0336] На фиг. 21C представлена иллюстративная экспрессия и показатели генного смещения для пептидов с тяжелой меткой, наблюдаемые в эксперименте с УФ-обработкой (красные точки, нанесенные на график в соответствии с экспрессией K562) по сравнению с меченными светом пептидами (серые точки, нанесенные на график в соответствии с экспрессией DC).
[0337] На фиг. 21D представлена иллюстративная диаграмма, отражающая перекрывание меченных тяжелой меткой генов, являющихся источниками пептидов, в соответствии с экспериментами в лизатах и с УФ-обработкой. Названия генов имеют цвет в соответствии с функциональным классом.
[0338] На фиг. 22A представлена иллюстративная блок-схема, отражающая протокол анализа, описанный в настоящем описании, для валидации стимулируемых HLA класса II CD4+T-клеток и T-клеточных ответов.
[0339] На фиг. 22B представлена иллюстративная схема конструкции димеров белков HLA для анализа пептидного обмена (верхняя панель) и графическое представление иллюстративного процесса анализа (нижняя панель). На фиг. представлена "10XHis" в качестве SEQ ID NO: 20.
[0340] На фиг. 23 представлено иллюстративное графическое представление иллюстративной конструкции вектора для экспрессии MHC-II для скрининга новых связывающих пептидов и обеспечения экспрессированного белкового продукта. На фиг. представлена SEQ ID NO: 39 и представлена "10XHis" в качестве SEQ ID NO: 20.
[0341] На фиг. 24 представлена иллюстративная блок-схема трансфекции, очистки и отщепления пептида-местозаполнителя от бета-цепи.
[0342] На фиг. 25A представлена иллюстративная графическая иллюстрация, демонстрирующая вектор, кодирующий пептиды CLIP, которые ассоциированы с повышенной секрецией экспрессируемых пептидов MHC-II. На фиг. представлены SEQ ID NO: 1 и 21 в порядке встречаемости.
[0343] На фиг. 25B представлено иллюстративное графическое представление более коротких и более длинных форм нуклеиновых кислот, кодирующих CLIP0 и CLIP1, соответственно.
[0344] На фиг. 25C представлен иллюстративный репрезентативный результат анализа геля с красителем Кумасси для альфа- и бета-цепей с более длинным clip или без него.
[0345] На фиг. 26A представлена иллюстративная графическая иллюстрация анализа TR-FRET.
[0346] На фиг. 26B представлены иллюстративные репрезентативные данные поляризации из анализа связывания пептидов HLA класса II с использованием анализа резонансного переноса энергии флуоресценции (FRET) с использованием определенных пептидов.
[0347] На фиг. 26C представлены иллюстративные репрезентативные данные поляризации из анализа связывания пептидов HLA класса II с использованием анализа резонансного переноса энергии флуоресценции (FRET) с использованием определенных пептидов.
[0348] На фиг. 26D представлено иллюстративное процентное вытеснение связанного с конструкцией MHC пептида, вычисленное из повышения флуоресценции.
[0349] На фиг. 26E представлено иллюстративное процентное вытеснение связанного с конструкцией MHC пептида, вычисленное из повышения флуоресценции.
[0350] На фиг. 26F представлен иллюстративный пептидный обмен посредством анализа с использованием дифференциальной сканирующей флуориметрии (DSF). Представлено графическое представление, демонстрирующее иллюстративный механизм детекции диссоциации пептида из MHC класса II при нагревании, которое также диссоциирует гетеродимер MHC класса II, что приводит к связыванию флуорофора и высокой флуоресценции. Также представлена иллюстративная схема вытеснения пептида-местозаполнителя посредством эпитопного пептида. Также представлены иллюстративные кривые плавления против температуры. На фиг. представлена "10XHis" в качестве SEQ ID NO: 20.
[0351] На фиг. 26G представлена иллюстративная растворимая конструкция HLA-DM и ее применение для проведения пептидного обмена для MHC класса II. Представленная конструкция содержит промотор CMV, кодирующую последовательность для бета-цепи HLA-DM и кодирующую последовательность для альфа-цепи HLA-DM ниже секреторной (лидерной) последовательности и последовательность BAP на 3'-конце кодирующей бета-цепь последовательности; His-метку на 3'-конце кодирующей альфа-цепь последовательности. Эти две цепи разделены встроенной последовательностью рибосомального пропускания. Конструкцию экспрессировали в клетках Expi-CHO и белок, секретируемый в культуральную среду, очищали.
[0352] На фиг. 26H представлены иллюстративные данные эксклюзионной хроматографии с использованием HLA-sDM для проведения пептидного обмена.
[0353] На фиг. 27A представлена иллюстративная графическая иллюстрация иллюстративной схемы репертуара тетрамеров DRB.
[0354] На фиг. 27B представлена иллюстративная графическая иллюстрация иллюстративной схемы репертуара тетрамеров класса II.
[0355] На фиг. 27C представлена иллюстративная графическая иллюстрация обобщения охвата репертуара тетрамеров DRB для аллеля DRB1 для пептидного обмена.
[0356] На фиг. 27D представлен иллюстративный охват продуцирования аллелей MHC класса II человека.
[0357] На фиг. 27E представлен иллюстративный результат окрашивания тетрамеров в образцах, индуцированных эпитопами Flu (ответ памяти) или эпитопами ВИЧ (наивный ответ).
[0358] На фиг. 28A представлено иллюстративное графическое представление способа оценки пептидов в отношении рестрикции HLA класса II посредством анализа флуоресцентной поляризации, который обеспечивает способ быстрой идентификации рестрикции аллелей для пептидов эпитопов. Принцип анализа, представленный на фиг. 28A, позволяет измерение аффинности и однозначное измерение пептидного обмена.
[0359] На фиг. 28B представлено иллюстративное обобщение множества исследованных условий анализа (верхняя панель) в анализе флуоресцентной поляризации с DRB1*01:01. Также представлена иллюстрация растворимого аллеля MHC класса II и полноразмерного аллеля MHC класса II с трансмембранным доменом в детергентной мицелле (нижняя панель), оба из которых были сконструированы с пептидом-местозаполнителем с расщепляемым линкером для применения в этом анализе.
[0360] На фиг. 28C представлено иллюстративное графическое представление анализов для исследования аллеля полноразмерного и растворимого белка, ранее показанных на фиг. 28B, нижняя панель. В кратком изложении, в клетках экспрессируют аллели как полноразмерных, так и растворимых белков. Мембраносвязанную полноразмерную форму белка аллеля собирают посредством пермеабилизации мембраны, в то время как секретируемую форму собирают из клеточного супернатанта. Собранные белки аллелей HLA класса II очищают, пропуская их через никелевые колонки (Ni2+).
[0361] На фиг. 28D представлены иллюстративные данные, демонстрирующие, что способ очистки не влияет на эффективность пептида. Слева представлены средние величины IC50 из экспериментов с использованием очищенного посредством L243 полоразмерного HLA-DR1 и очищенного посредством Ni2+ полноразмерного HLA-DR1.
[0362] На фиг. 28E представлены иллюстративные данные, демонстрирующие, что выбор растворимой формы (sDR1) или полноразмерной формы (fDR1) не влияет на эффективность пептида. Слева представлены средние значения IC50 из экспериментов с использованием формы sDR1 или fDR1. FP, флуоресцентная поляризация.
[0363] На фиг. 28F представлено иллюстративное графическое изображение иллюстративной оценки спрогнозированных пептидов посредством neonmhc2 и NetMHCIIpan в анализе связывания и идентификации дискордантных пептидов.
[0364] На фиг. 28G представлены иллюстративные данные скрининга посредством флуоресцентной поляризации для оценки спрогнозированных посредством neonmhc2 пептидов; также в качестве тепловой карты показано процентное ингибирование связывания зонда для каждой использованной концентрации пептида. Зеленым цветом представлено хорошее связывание, которое пропорционально интенсивности цвета. Желтым цветом представлено промежуточное связывание, и красным цветом представлено плохое связывание, что также указано соответствующими величинами процентного ингибирования.
[0365] На фиг. 28H представлено обобщение оценки спрогнозированных посредством neonmhc2 пептидов в иллюстративном анализе связывания.
[0366] На фиг. 29 представлено иллюстративное среднее количество пептидов из средней экспериментальной реплики MAPTAC™ (50 миллионов клеток), на каждый аллель HLA.
[0367] На фиг. 30A-30C представлен иллюстративный анализ центра связывания для аллелей HLA класса II MAPTAC™ +/- HLA-DM и точность мультиаллельной обратной свертки. На фиг. 30A представлены иллюстративные логотипы последовательностей для одного репрезентативного аллеля HLA-DR, -DQ и -DP в соответствии с MAPTAC™ с сотрансфекцией HLA-DM и без нее (клеточная линия expi293) и IEDB, где высота каждой аминокислоты пропорциональна ее частоте. Аминокислоты с частотой более 10% представлены цветом в зависимости от химических свойств; все другие представлены серым цветом. Пептиды выравнивают в соответствии с инструментом GibbsCluster (дополнительные способы), и логотипы соответствуют всем пептидам, включая пептиды, которые не соответствуют близко общему мотиву (например, никакие пептиды не помещаются в кластер "мусор"). На фиг. 30B представлено иллюстративное описание распределений на кластеры для пептидов MAPTAC™ (20 на аллель), внесенных в наборы данных MS для общего DR. Наборы данных подвергали обратной свертке с использованием GibbsCluster. Каждая окрашенная рамка соответствует одному пептиду MAPTAC™. Цвет рамки указывает на то, к какому кластеру он отнесен, и серые столбики указывают на то, из какого аллеля пептид произошел. На фиг. 30C представлен иллюстративный график, показывающий долю пептидов, демонстрирующих 0, 1, 2, 3 или 4 ожидаемых остатков в якорных положениях для аллелей, представленных на фиг. 30B. Якорные положения были определены как четыре положения с наиболее низкой энтропией и "ожидаемые" остатки были определены как остатки с частотой ≥10% в этих положениях.
[0368] На фиг. 31A-31F представлена иллюстративная архитектура и сопоставительный анализ алгоритма прогнозирования связывания neonmhc2. На фиг. 31A представлена иллюстративная архитектура сверточной нейронной сети (CNN), обученной различать моноаллельные пептиды HLA класса II и перемешенные совпадающие по длине ловушки. На схеме указано использование слоя вложения признаков аминокислот, 2 сверточных слоев шириной 6, присутствие соединений "прыжок до конца" и комбинация операции среднего и максимального пулинга в качестве ввода в конечный логистический выводной узел. На фиг. 31B представлена иллюстративная положительная прогностическая величина (PPV) для NetMHCIIpan и neonmhc2, оцененная для распределения данных MAPTAC™, которые не использовались для обучения или гиперпараметрической оптимизации. Для каждого аллеля, n выявленных посредством MS пептидов оценивали совместно с 19n совпадающих по длине ловушек, взятых из того же набора генов-источников, и каждый из n пептидов средства прогноза с наилучшим рангом (например, лучшие 5%) называли положительным результатом. В соответствии с этим протоколом оценки, PPV идентична отзыву, поскольку количество ложноположительных результатов и ложноотрицательных результатов обязательно является равным. На фиг. 31C представлена иллюстративная PPV для NetMHCIIpan и neonmhc2 на наборе данных TGEM. Для каждого аллеля n пептидов с наилучшим рангом называли положительными результатами, где n представляет собой количество подтвержденных иммуногенных эптопов в оцененном наборе. На фиг. 31D представлены иллюстративные результаты индукции T-клеток ex vivo для неоантигенных пептидов. Селекцию пептидов проводили на основе высоких показателей neonmhc2 и слабых показателей NetMHCIIpan для HLA-DRB1*11:01. На фиг. представлены SEQ ID NO: 87-89, 91, 90, 2, 92-94, 3 и 95-96, соответственно, в порядке встречаемостир. На фиг. 31E представлено сравнение моделей, обученных на моноаллельных данных MAPTAC, против подвергнутых обратной свертке мультиаллельных данных, оцененных на удержанных моноаллельных данных. Значения представлены для neonmhc2, где обучающий набор данных подвергнут уменьшению выборки, чтобы соответствовать размеру обучающего набора для обратной свертки. На фиг. 31F представлена PPV для набора данных TGEM для NetMHCIIpan-v3.1, обученного посредством обратной свертки средства прогноза, и neonmhc2 (с уменьшением выборки и без нее). Для каждого аллеля n пептидов с наилучшим рангом были названы положительными результатами, где n представляет собой количество подтвержденных иммуногенных эпитопов в оцененной выборке.
[0369] На фиг. 32A-32E представлено иллюстративное представление генов и процессинга белков в пептидомах опухолей HLA класса II. На фиг. 32A представлены иллюстративные результаты выявленного против ожидаемого количества пептидов HLA класса II на ген при определении посредством совместного анализа наборов данных для рака ободочной и прямой кишки, меланомы и рака яичника. Ожидаемое количество получено путем умножения длины генов на уровень экспрессии. Ожидаемые и выявленные количества суммировали для соответствующих образцов. Гены с известным присутствием в плазме обозначены в соответствии с их концентрацией. На фиг. 32B представлены иллюстративные результаты ожидаемой против наблюдаемой частоты пептидов в соответствии с клеточной локализацией. На фиг. 32C представлены иллюстративные результаты распределения показателей увеличения представленности (соотношение выявленных и ожидаемых наблюдений, как и на части фиг. 32B) для генов, регулируемых протеасомой. Наборы генов включают гены с известными участками убиквитинилирования и гены, содержание продуктов которых возрастает при применении ингибитора протеасом. На фиг. 32D представлена диаграмма, показывающая три иллюстративных рабочих модели для того, как пептиды HLA класса II процессируются, в соответствии с которой i) катепсины и другие ферменты расщепляют белки на пептидные фрагменты, которые затем связываются посредством HLA, ii) белки или несвернутые полипептиды связывают HLA, а затем расщепляются до пептидной длины, iii) белки частично расщепляются перед связыванием и далее укорачиваются после связывания. Каждая модель соответствует отличающемуся подходу прогнозирования. На фиг. 32E представлено абсолютное увеличение PPV, наблюдаемое для логистических регрессионных моделей, которые включали связанные с процессингом переменные, и прогнозов связывания neonmhc2, по сравнению с моделями, в которых использовались только прогнозы связывания. Оценку проводили для одиннадцати образцов, которые подвергали определению профиля посредством антитела против HLA-DR (те же образцы, которые анализировали для фиг. 30B); каждая точка соответствует одному образцу. Звездочки обозначают значимое улучшение (*: p<0,01, **: p<0,001, ***: p<0,0001) в соответствии с двухсторонним парным t-критерием. Тот же анализ представлен на фиг. 40B однако вместо этого на нем использовался NetMHCIIpan в качестве основного средства прогнозирования. Способы выбора ловушек и вычисления PPV идентичны способам, использованным для фиг. 31B.
[0370] На фиг. 33A-33G представлены иллюстративные результаты идентификации и прогнозирования опухолевых антигенов, презентируемых дендритными клетками. На фиг. 33A представлено иллюстративное графическое представление экспериментального процесса для идентификации DC-презентируемых лигандов HLA-II, происходящих из злокачественных клеток (K562). Злокачественные клетки выращивали в среде SILAC до полного включения, либо лизировали, либо облучали, а затем сеяли дендритные клетки моноцитарного происхождения. Презентируемые пептиды выделяли посредством общего антитела против DR и секвенировали посредством LC-MS/MS. На фиг. 33B представлены иллюстративные данные, отражающие эффективность прогнозирования для происходящих из опухоли пептидов, презентируемых дендритными клетками, с использованием того же соотношения наиболее подходящей последовательности и ловушки и показателей эффективности, что и на фиг. 21A. Эффективность представлена для моделей на основе NetMHCIIpan и neonmhc2 с использованием и без использования признаков процессинга. На фиг. 33C представлено иллюстративное распределение экспрессии генов для генов-источников для меченных тяжелой меткой пептидов, выявленных в эксперименте с УФ-обработкой (красная кривая, нанесенная в соответствии с экспрессией K562) по сравнению с генами-источниками меченных легкой меткой пептидов (серая кривая, построенная в соответствии с экспрессией DC). На фиг. 33D представлен иллюстративный график PPV при соотношении наиболее подходящей последовательности и ловушки 1:499 для прогнозирования презентируемых опухолевых антигенов с использованием моделей на основе NetMHCIIpan и neonmhc2 с признаками процессинга или без них. Точки данных слева направо соответствуют образцам: клетки донора 1, обработанные HOCl: непрерывная экспрессия NetMHCIIpan; непрерывная экспрессия NetMHCIIpan + генное смещение; непрерывная экспрессия NetMHCIIpan + генное смещение + перекрывание DQ, режим полного процессинга; клетки донора 1, обработанные УФ-излучением: neonmhc2; neonmhc2 + пороговая экспрессия; neonmhc2 + непрерывная экспрессия; neonmhc2 + непрерывная экспрессия + генное смещение; neonmhc2 + непрерывная экспрессия + генное смещение + перекрывание DQ. На фиг. 33E представлено значение различных локализаций генов и функциональных классов при прогнозировании тяжелых (происходящих из K562) и легких (происходящих из DC) пептидов, соответственно. Значения P вычислены в соответствии с логистической регрессией, которая учитывает показатель связывания neonmhc2 и экспрессию гена-источника. Цвета столбиков указывают на знак, ассоциированный с коэффициентом регрессии. На фиг. 33F представлено иллюстративное графическое представление результатов, демонстрирующих перекрывание генов-источников для происходящих из опухолевых клеток пептидов (окрашенных в зависимости от функционального класса) в экспериментах с обработкой УФ-излучением и HOCl. На фиг. 33G представлены иллюстративные данные, демонстрирующие PPV для прогнозирования презентируемых опухолевых антигенов у второго донора с использованием логистических моделей, аппроксимированных для пептидов с тяжелой меткой, выявленных у первого донора. Аппроксмацию моделей проводили с использованием neonmhc2 для только связывания; связывания и экспрессии; или связывания, экспрессии и бинарной переменной, указывающей на то, происходил ли пептид из митохондриального гена.
[0371] На фиг. 34A-34B представлена иллюстративная охарактеризация данных MAPTAC™, связанных с фиг. 29. На фиг. 34A представлен иллюстративный анализ HLA клеточной поверхности посредством FACS в клеточных линиях Expi293, трансфицированных конструкциями MAPTAC™, кодирующими меченный аффинной меткой HLA-A*02:01-BAP. На фиг. 34B представлен иллюстративный анализ HLA клеточной поверхности посредством FACS в клеточных линиях Expi293, трансфицированных конструкциями MAPTAC™, кодирующими меченный аффинной меткой HLA-DRB1*11:01-BAP (снизу). Экспрессию HLA на клеточной поверхности в трансфицированных клетках Expi293 (оранжевый) сравнивали с окрашенными нетрансфицированными Expi293 (синий), неокрашенными нетрансфицированными Expi293 (красный), окрашенными PBMC (темно-зеленый) и неокрашенными PBMC (светло-зеленый). В качестве красителя для HLA класса I использовался W6/32 (общий для HLA класса I), в то время как в качестве красителя для HLA класса II использовался REA332 (общий для HLA класса II).
[0372] На фиг. 35 представлено иллюстративное сравнение логотипов MAPTAC™ и IEDB, связанное с фиг. 30A. Представлена измеренная и спрогнозированная посредством NetMHCIIpan аффинность для выявленных посредством MS пептидов, которые не имели высоких показателей NetMHCIIpan, но хорошо поддерживались MS (оцененная интенсивность пика >70 и размер вложенного множества ≥1).
[0373] На фиг. 36A-36C представлен иллюстративный анализ точности данных MAPTAC™ для HLA-DR1, связанный с фиг. 30A-30C. На фиг. 36A представлены иллюстративные показатели NetMHCIIpan3.1 для пептидов HLA-DR1 MAPTAC™ (зеленый) (длины 12-23) по сравнению с 50000 совпадающими по длине пептидами-ловушками, случайным образом отобранными из протеома (синий), для распространенных аллелей. На фиг. 36B представленная иллюстративная измеренная и спрогнозированная посредством NetMHCIIpan аффинность для иллюстративных наблюдаемых посредством MS пептидов, которые не демонстрировали высокие показатели NetMHCIIpan, но хорошо поддерживались MS (оцененная интенсивность пика >70 и размер вложенного множества ≥1). На фиг. представлены SEQ ID NO: 40-86, сверху вниз, слева направо, соответственно, в порядке встречаемости. На фиг. 36C представлены иллюстративные логотипы последовательностей HLA класса II для аллелей HLA-DRB1, определенных посредством MAPTAC™ в различных типах клеток.
[0374] На фиг. 37A-37C представлен дополнительный иллюстративный анализ мотивов MAPTAC™, связанный с фиг. 30A-30C. На фиг. 37A представлены логотипы последовательностей полученные посредством MAPTAC™ для экспериментов с сотрансфекцией HLA-DM и без нее (клеточная линия expi293). На фиг. 37B представлены логотипы последовательностей для нескольких аллелей HLA класса I согласно MAPTAC™ и IEDB. Следует отметить, что A*32:01 не демонстрирует высокую частоту Q в P2 и C*03:03 не демонстрирует высокую частоту Y в P9, что отличается от предшествующих исследований, в которых использовалась мультиаллельная обратная свертка; логотип для B*52:01 ранее не был опубликован. На фиг. 37C представлено иллюстративное выравнивание выявленных посредством MAPAC™ пептидов с последовательностью гена CD74.
[0375] На фиг. 38Ai-38D представлена иллюстративная статистика эффективности neonmhc2 и проточного окрашивания T-клеток, связанная с фиг. 31A-31D. На фиг. 38Ai представлена иллюстративная эффективность neonmhc2 в зависимости от размера обучающей выборки данных. PPV вычисляли аналогично и с использованием тех же пептидов для оценки, что и на фиг. 31B; однако обучающие данные случайным образом подвергали уменьшению выборки для имитации меньших обучающих наборов данных. На фиг. 38Aii представлены иллюстративные логотипы последовательностей для пептидных кластеров, происходящих из мультиаллельного лигандома HLA-DR с использованием GibbsCluster (параметры по умолчанию; кластер "мусор" разрешен). На фиг. 38B представлены иллюстративные репрезентативные графики проточной цитометрии для экспрессии IFN-γ клетками CD4+ из индуцированных образцов, отобранных с использованием неоантигенных пептидов, спрогнозированных посредством neonmhc2. Дельта-величины вычисляли путем вычитания процента CD4+ клеток, экспрессирующих IFN-γ, при селекции с использованием неоантигена (+пептид), из процента CD4+ клеток, экспрессирующих IFN-γ, при селекции в отсутствии неоантигена (без пептида). Левые два графика являются репрезентативными для неоантигена, который индуцировал ответ CD4+ T-клеток (PEASLYGALSKGSGG (SEQ ID NO: 2)) и неоантигена, который не индуцировал ответ T-клеток (PATYILILKEFCLVG (SEQ ID NO: 3)). На фиг. 38C представлены иллюстративные дельта-величины для лунок с единичными неоантигенными пептидами neonmch2. Пептиды считались наилучшими индуцирующими пептидами, если они имели положительный ответ (дельта-ответ выше 3%, выделены цветом). На фиг. представлены SEQ ID NO: 87-91, 2, 92-94, 3 и 95-96, соответственно, в порядке встречаемости. На фиг. 38D представлены иллюстративные логотипы последовательностей для кластеров, полученных для мультиаллельных лигандомов HLA-DR с использованием GibbsCluster (параметры по умолчанию; кластер "мусор" разрешен).
[0376] На фиг. 39A-39C представлен дополнительный анализ клеток-источников для HLA класса II, связанный с фиг. 32A-32E. На фиг. 39A представлены иллюстративные процентные ранжированные показатели neonmhc2 для пептидов HLA класса II, выявленные в 4 образцах PBMC, подвергнутых определению профиля посредством общего антитела против DR (RG1248, RG1104, RG1095 и HDSC из фиг. 30B), в соответствии с тем, присутствует ли ген-источник пептида в плазме человека. Для каждого пептида использовали наилучший (наиболее низкий) процентный ранг среди аллелей, присутствующих у донора. Для сравнения показаны показатели для случайных совпадающих по длине протеомных ловушек. Блочные диаграммы указывают на 5-й, 25-й, 50-й, 75-й и 95-й перцентили. На фиг. 39B представлены иллюстративные количества выявленных против ожидаемых пептидов на ген для HLA класса I с использованием той же методологии, что и на фиг. 32A. Данные соответствуют тем же типам опухоли (ободочной и прямой кишки, яичника и меланома). Гены, присутствующие в плазме человека, выделены синим цветом и размер для них соответствует их концентрации. На фиг. 39C представлена иллюстративная относительная согласованность наблюдения для пептидов в отношении двух различных профилей экспрессии генов. Для каждого образца количества пептидов на генном уровне моделировали в качестве линейной комбинации профиля совокупной экспрессии генов в опухоли и экспрессии генов профессиональными APC. Соотношение коэффициентов определяет относительную согласованность каждого профиля экспрессии с репертуаром пептидов. Планки погрешности соответствуют 95% доверительному интервалу, вычисленному посредством бустрэп-перевыборки.
[0377] На фиг. 40A-40B представлен дополнительный иллюстративный анализ мотивов процессинга, связанный с фиг. 32A-32E. На фиг. 40A представлены иллюстративные частоты аминокислот вблизи участков отщепления N-концевых и C-концевых пептидов относительно средних протеомных частот (применимо для вышележащих положений U3-U1 и нижележащих положений D1-D3) или относительно средних пептидных частот (применимо для внутренних положений N1-C1), наблюдаемых в донорских PBMC, происходящих из моноцитов дендритных клетках, клетках рака ободочной и прямой кишки, меланоме, рак яичника и клеточной линии expi293 (используемой для получения большинства данных MAPTAC™). На фиг. 40B представлен тот же анализ, что и на фиг. 32E, но с использованием NetMHCIIpan в качестве основного средства прогноза. Абсолютное повышение PPV наблюдалось для логистических регрессионных моделей, которые включали связанные с процессингом переменные в дополнение к прогнозам NetMHCIIpan (по сравнению с моделями только с NetMHCIIpan), для восьми образцов, подвергнутых определению профиля посредством антител против HLA-DR (те же образцы, которые анализировались для фиг. 31B). Звездочкой указано значимое улучшение (*: p<0,01, **: p<0,001, ***: p<0,0001) в соответствии с двухсторонними парными t-критериями.
[0378] На фиг. 41 представлена иллюстративная система наименований, использованная для указания на положения выше пептидов, в пептидах и ниже пептидов.
[0379] На фиг. 42A представлена диаграмма, на которой показан иллюстративный процесс анализа эндогенно процессируемых и презентируемых посредством HLA-1 и HLA класса II пептидов с использованием nLC-MS/MS.
[0380] На фиг. 42B представлен график, демонстрирующий иллюстративные результаты эксперимента из анализа посредством nLC-MS/MS триптических пептидов с или без FAIMS. Также представлено репрезентативное перекрывание при детекции пептидов HLA-1 и HLA класса II посредством анализа nLC-MS/MS с или без FAIMS в указанном масштабе анализа.
[0381] На фиг. 43A представлена иллюстративная кислотная и основная обращенно-фазовая детекция фракционированных пептидов для HLA класса I с или без FAIMS.
[0382] На фиг. 43B представлены иллюстративные экспериментальные результаты, демонстрирующие детекцию связываемых HLA класса I уникальных пептидов, нанесенные на график против времени удержания.
[0383] На фиг. 44A представлена иллюстративная кислотная и основная обращенно-фазовая детекция фракционированных пептидов для HLA класса II с или без FAIMS.
[0384] На фиг. 44B представлены иллюстративные экспериментальные результаты, демонстрирующие детекцию связываемых HLA класса II уникальных пептидов, нанесенные на график против времени удержания.
[0385] На фиг. 45 представлен иллюстративный график размера пересечения связывающих пептидов HLA класса I, выявленных с использованием указанных способов (слева), и диаграмма Венна для иллюстративного стандартного процесса и оптимизированного процесса детекции посредством LC-MS/MS связывающих пептидов HLA класса I (справа).
[0386] На фиг. 46 представлен иллюстративный график размера пересечения связывающих пептидов HLA класса II, выявленных с использованием указанных способов (слева), и диаграмма Венна для иллюстративного стандартного процесса и оптимизированного процесса детекции посредством LC-MS/MS связывающих пептидов HLA класса II (справа).
ПОДРОБНОЕ ОПИСАНИЕ
[0387] Подразумевается, что все термины следует понимать, как их понимает специалист в данной области. Если не определено иначе, все технические и научные термины, используемые в настоящем описании, обладают тем же значением, которое обычно подразумевает специалист в области, к которой относится изобретение.
[0388] Заголовки разделов, используемые в настоящем описании, приведены только для организационных целей, и их не следует истолковывать как ограничивающие описанный объект.
[0389] Хотя различные признаки настоящего изобретения могут быть описаны в контексте единичных вариантов осуществления, признаки также могут быть предоставлены по отдельности или в любой подходящей комбинации. Напротив, хотя для ясности настоящее изобретение может быть описано в настоящем описании в контексте раздельных вариантов осуществления, изобретение также может быть осуществлено в едином варианте осуществления.
[0390] Настоящее изобретение основано на важном открытии, что презентация антигенов, в частности, антигенов злокачественной опухоли, посредством специфических пар альфа- и бета-цепей HLA класса II может быть спрогнозирована с высокой степенью достоверности с использованием новой компьютерной машинно-обучаемой модели для прогнозирования презентации пептидов HLA, которая позволяет использовать специфические для HLA класса II пептиды для усовершенствования иммунотерапии.
[0391] В одном аспекте настоящее изобретение относится к способу прогнозирования пептидов, которые точно могут образовывать пару или связывать определенный гетеродимер альфа- и бета-цепей HLA класса II, так что высокоточное связывание пептида с белком HLA класса II (содержащим гетеродимер альфа- и бета-цепей) обеспечивает презентацию специфичного к нему пептида T-лимфоцитам, тем самым индуцируя специфический иммунный ответ и избегая какой-либо перекрестной реактивности или иммунной неразборчивости. Несколько последних исследований показали, что CD4+ T-клетки также могут распознавать презентируемые посредством HLA класса II лиганды и участвовать в контроле опухоли. В вакцинах против злокачественной опухоли и других иммунотерапевтических средствах в идеале используется нацеливание ответов CD4+ T-клеток, однако текущие усилия полностью упускают прогнозирование антигенов HLA класса II, поскольку точность современных инструментов прогнозирования является недостаточной.
[0392] В одном аспекте настоящее изобретение относится к способу прогнозирования пептидов, которые точно могут связываться с определенным белком HLA класса II, так что пептид может активировать более длительный и устойчивый иммунный ответ, когда пептид вводят терапевтически индивидууму, у которого экспрессируется определенный собственный белок HLA класса II, посредством способности белка HLA класса II активировать CD4+ T-клетки и стимулировать иммунологическую память. В некоторых вариантах осуществления способ, описанный в настоящем описании, обеспечивает улучшение специфического прогнозирования для белков HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 1,1 раза прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогноза. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 2 раза прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 3 раза прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 4 раза прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 5 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 6 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 7 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 8 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 9 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 10 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 15 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 20 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 30 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 40 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 50 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования. В некоторых вариантах осуществления способ, описанный в настоящем описании, демонстрирует улучшение по меньшей мере приблизительно в 60 раз прогнозирования для определенного белка HLA класса II относительно доступного в настоящее время средства прогнозирования.
[0393] В одном аспекте в настоящем описании описаны способы иммунотерапии, приспособленной или персонализированной для конкретного индивидуума. У каждого индивидуума или пациента экспрессируется определенный набор белков HLA класса I и HLA класса II. HLA-типирование является хорошо известным способом, который позволяет определение конкретного репертуара белков HLA, экспрессируемых у индивидуума. После того, как установления гетеродимеров HLA, экспрессируемых у конкретного индивидуума, наличие усовершенствованного, продвинутого и надежного способа, как описано в настоящем описании, для прогнозирования пептидов, которые могут связываться с конкретным гетеродимером альфа-цепи и бета-цепи HLA класса II с высокой точностью, может гарантировать, что может быть индуцирован конкретный иммунный ответ, адаптированный конкретно для данного индивидуума.
[0394] В настоящем описании использование формы единственного числа включает множественное число, если контекст не указывает на иное. Необходимо отметить, что, как используют в настоящем описании объекты в форме единственного числа включают множественное число упоминаемых объектов, если контекст явно не указывает на иное. В настоящей заявке использование "или" означает "и/или", если не указано иное. Более того, использование термина "включающий", а также других форм, таких как "включать", "включает" и "включенный" не является ограничивающим. Термины "один или более" или "по меньшей мере один", такие как одни или более или по меньшей мере один представитель(и) из группы представителей, ясен сам по себе, для дальнейшего пояснения, этот термин охватывает, среди прочих, указание на любой один из указанных представителей или на любые два или более из указанных представителей, как например, любые ≥3, ≥4, ≥5, ≥6 или ≥7 и т.д. из указанных представителей и вплоть до всех из указанных представителей.
[0395] Указание в описании на "некоторые варианты осуществления", "вариант осуществления" "один вариант осуществления" или "другие вариант осуществления" означает, что признак, структура или характеристика, описанные в связи с вариантами осуществления, включены по меньшей мере в некоторые варианты осуществления, но не обязательно во все варианты осуществления, настоящего изобретения.
[0396] Как используют в настоящем описании и формуле(пунктах формулы) изобретения, слова "содержащий" (и любая форма слова "содержащий", такая как "содержать" и "содержит"), "имеющий" (и любая форма слова "имеющий", такая как "иметь" и "имеет"), "включающий" (и любая форма слова "включающий", такая как "включает" и "включать") или "вмещающий" (и любая форма слова "вмещающий", такая как "вмещает" и "вмещать") являются включающими или открытыми и не исключают дополнительных не указанных элементов или стадий способа. Предусматривается, что любой вариант осуществления, описанный в настоящем описании, может быть применен в отношении любого способа или композиции по изобретению и наоборот. Более того, композиции по изобретению можно использовать для выполнения способов по изобретению.
[0397] Подразумевают, что термин "приблизительно" или "примерно", как используют в рамках изобретения при указании на поддающуюся измерению величину, такую как параметр, количество, период времени и т.п., охватывает отклонение +/-20% или менее, +/-10% или менее, +/-5% или менее, или +/-1% или менее указанной величины и от указанной величины, при условии, что такие отклонения являются приемлемыми для осуществления настоящего изобретения. Следует понимать, что величина, к которой относится определение "приблизительно" или "примерно", сама по себе также конкретно раскрыта.
[0398] Термин "иммунный ответ" включает опосредуемый T-клетками и/или опосредуемый B-клетками иммунный ответ, на который влияет модулирование костимуляции T-клеток. Иллюстративные иммунные ответы включают T-клеточные ответы, например, продуцирование цитокинов и клеточную цитотоксичность. Кроме того, термин "иммунный ответ" включает иммунный ответ, на который непрямо влияет активация T-клеток, например, продуцирование антител (гуморальные ответы), и активация отвечающих на цитокины клеток, например, макрофагов.
[0399] Под "рецептором" подразумевают биологическую молекулу или группу молекул, способных связывать лиганд. Рецептор может служить для передачи информации в клетке, клеточном образовании или организме. Рецептор включает по меньшей мере один рецепторный элемент и может содержать два или более рецепторных элементов, где каждый рецепторный элемент может состоять из белковой молекулы, например, молекулы гликопротеина. Рецептор имеет структуру, которая комплементирует структуру лиганда и может образовывать комплекс с лигандом в качестве партнера по связыванию. Сигнальная информация может передаваться посредством конформационных изменений рецептора после связывания с лигандом на поверхности клетки. В соответствии с настоящим изобретением, рецептор может относиться к белкам MHC классов I и II, способным образовывать комплекс рецептор/лиганд с лигандом, например, пептидом или пептидным фрагментом подходящей длины. Пептиды MHC класса I и класса II, которые кодируются аллелями HLA класса I и класса II, часто упоминаются в настоящем описании как пептиды HLA класса I и HLA класса II, соответственно, или пептиды HLA класса I и HLA класса II, или белки HLA класса I и класса II, или белки HLA класса I и HLA класса II, или молекулы HLA класса I и класса II, или их подобные распространенные варианты, как хорошо понятно в контексте обсуждения специалисту в данной области.
[0400] "Лиганд" представляет собой молекулу, которая способна образовывать комплекс с рецептором. В соответствии с настоящим изобретением, под лигандом подразумевают, например, пептид или пептидный фрагмент, который имеет подходящую длину и подходящие связывающие мотивы в его аминокислотной последовательности, так что пептид или пептидный фрагмент способен связываться и образовывать комплекс с белками MHC класса I или MHC класса II (т.е. белками HLA класса I и HLA класса II).
[0401] "Антиген" представляет собой молекулу, которая способна стимулировать иммунный ответ и которая может продуцироваться злокачественными клетками или инфекционными агентами, или при аутоиммунном заболевании. Антигены, распознаваемые T-клетками, как хелперными T-лимфоцитами (T-хелперные (TH) клетки), так и цитотоксическими T-лимфоцитами (CTL), распознаются не в качестве интактных белков, а скорее в качестве небольших пептидов, связанных с белками HLA класса I или класса II на поверхности клеток. В ходе естественного иммунного ответа антигены, которые распознаются связанными с молекулами HLA класса II на антигенпредставляющих клетках (APC), приобретаются извне клетки, интернализуются и процессируются в небольшие пептиды, которые связаны с молекулами HLA класса II. APC также могут перекрестно презентировать пептидные антигены посредством процессинга экзогенных антигенов и презентации процессированных антигенов на молекулах HLA класса I. Антигены, которые дают начало пептидам, которые распознаются связанными с молекулами MHC, HLA класса I, как правило, представляют собой пептиды, которые продуцируются в клетках, и эти антигены процессируются и связываются с молекулами MHC класса I. В настоящее время понятно, что пептиды, которые связываются с данными молекулами HLA класса I или класса II, характеризуются наличием общего связывающего мотива, и связывающие мотивы для большого количества различных молекул HLA класса I и II определены. Также можно синтезировать синтетические пептиды, которые соответствуют аминокислотной последовательности данного антигена и которые содержат связывающий мотив для данной молекулы HLA класса I или II. Затем эти пептиды можно добавлять к соответствующим APC, и APC могут использоваться для стимуляции ответа T-хелперных клеток или ответа CTL либо in vitro, либо in vivo. Связывающие мотивы, способы синтеза пептидов и способы стимуляции ответа T-хелперных клеток или ответа CTL известны и хорошо доступны специалисту в данной области.
[0402] Термин "пептид" используют взаимозаменяемо с терминами "мутантный пептид" и "неоантигенный пептид" в настоящем описании. Аналогично, термин "полипептид" используют взаимозаменяемо с "мутантным полипептидом" и "неоантигенным полипептидом" в настоящем описании. Под "неоантигеном" или "неоэпитопом" подразумевают класс опухолевых антигенов или опухолевых эпитопов, которые появляются в результате опухолеспецифических мутаций в экспрессируемом белке. Кроме того, настоящее изобретение относится к пептидам, которые содержат опухолеспецифические мутации, пептидам, которые содержат известные опухолеспецифические мутации, и мутантным полипептидам или их фрагментам, идентифицированным способом по настоящему изобретению. Эти пептиды и полипептиды называют в настоящем описании "неоантиенными пептидами" или "неоантигенными полипептидами". Полипептиды или пептиды могут иметь различную длину, могут быть либо в их природной (незаряженной) форме, либо в форме солей, и либо свободны от модификаций, таких как гликозилирование, окисление боковых цепей, фосфорилирование или любая посттрансляционная модификация, либо содержат эти модификации при условии, что данная модификация на нарушает биологическую активность полипептидов, как описано в настоящем описании. В некоторых вариантах осуществления неоантигенные пептиды по настоящему изобретению могут включать: для HLA класса I, 22 остатка в длину или менее, например, от приблизительно 8 до приблизительно 22 остатков, от приблизительно 8 до приблизительно 15 остатков, или 9 или 10 остатков; для HLA класса II, 40 остатков в длину или менее, например, от приблизительно 8 до приблизительно 40 остатков, от приблизительно 8 до приблизительно 24 остатков, от приблизительно 12 до приблизительно 19 остатков, или от приблизительно 14 до приблизительно 18 остатков. В некоторых вариантах осуществления неоантигенный пептид или неоантигенный полипептид содержит неоэпитоп.
[0403] Термин "эпитоп" включает любую белковую детерминанту, способную специфически связываться с антителом, антительным пептидом и/или антитело-подобной молекулой (включая, но не ограничиваясь ими, T-клеточный рецептор), как определено в настоящем описании. Эпитопные детерминанты, как правило, состоят из химически активных групп молекул, таких как аминокислоты или боковые цепи сахаров, и, как правило, имеют конкретные трехмерные структурные характеристики, а также конкретные зарядовые характеристики.
[0404] "T-клеточный эпитоп" представляет собой пептидную последовательность, которая может связываться молекулами MHC класса I или II в форме презентирующей пептид молекулы MHC или комплекса MHC, а затем в этой форме может распознаваться и связываться цитотоксическими T-лимфоцитами или T-хелперными клетками, соответственно.
[0405] Термин "антитело", как используют в рамках изобретения, включает IgG (включая IgGl, IgG2, IgG3 и IgG4), IgA (включая IgAl и IgA2), IgD, IgE, IgM и IgY, и подразумевается, что он включает целые, в том числе одноцепочечные целые, антитела и их антигенсвязывающие (Fab) фрагменты. Антигенсвязывающие фрагменты антител включают, но не ограничиваются ими, Fab, Fab' и F(ab')2, Fd (состоящий из VH и CH1), одноцепочечный вариабельный фрагмент (scFv), одноцепочечные антитела, связанный дисульфидной связью вариабельный фрагмент (dsFv) и фрагменты, содержащие либо домен VL, либо домен VH. Антитела могут быть животного происхождения. Антигенсвязывающие фрагменты антител, включая одноцепочечные антитела, могут содержать вариабельную область(и) отдельно или в комбинации с целым или частью следующих: шарнирная область, домены CH1, CH2 и CH3. Также включены любые комбинации вариабельной области(ей) и шарнирной области, доменов CH1, CH2 и CH3. Антитела могут представлять собой моноклональные, поликлональные, химерные, гуманизированные и человеческие моноклональные и поликлональные антитела, которые, например, специфически связывают ассоциированный с HLA полипептид или комплекс HLA-связывающий HLA пептид (HLA-пептид). Специалисту в данной области будет понятно, что различные иммуноаффинные способы являются пригодными для увеличения в количестве растворимых белков, таких как растворимые комплексы HLA-пептид или мембраносвязанные ассоциированные с HLA полипептиды, например, которые протеолитически отщепляются от мембраны. Они включают способы, в которых (1) одно или более антител, способных специфически связываться с растворимым белком, иммобилизуют на фиксированной или подвижной подложке (например, пластмассовые лунки или гранулы из смолы, латексные или парамагнитные гранулы), и (2) раствор, содержащий растворимый белок из биологического образца, пропускают над покрытой антителом подложкой, позволяя растворимому белку связаться с антителами. Подложку с антителом и связанным растворимым белком отделяют от раствора и необязательно антитело и растворимый белок диссоциируют, например, посредством варьирования pH, и/или ионной силы, и/или ионной композиции раствора, в котором находятся антитела. Альтернативно можно использовать способы иммунопреципитации, в которых антитело и растворимый белок комбинируют и позволяют им образовывать макромолекулярные агрегаты. Макромолекулярные агрегаты могут быть отделены от раствора эксклюзионными способами или посредством центрифугирования.
[0406] Термин "иммунная очистка (IP)" (или иммуноаффинная очистка или иммунопреципитация) представляет собой способ, хорошо известный в данной области и широко используемый для выделения желаемого антигена из образца. Как правило, способ вовлекает приведение в контакт образца, содержащего желаемый антиген, с аффинным матриксом, содержащим антитело к антигену, ковалентно связанное с твердой фазой. Антиген в образце связывается с аффинным матриксом через иммунохимическую связь. Затем аффинный матрикс промывают для удаления каких-либо несвязанных компонентов. Антиген извлекают из аффинного матрикса путем изменения химического состава раствора в контакте с аффинной матрицей. Иммунную очистку можно проводить на колонке, содержащей аффинный матрикс, в случае которого раствор представляет собой элюент. Альтернативно иммунная очистка может представлять собой периодический процесс, в случае которого аффинный матрикс поддерживают в качестве суспензии в растворе. Важной стадией в этом процессе является удаление антигена из матрикса. Этого обычно достигают путем увеличения ионной силы раствора в контакте с аффинным матриксом, например, посредством добавления неорганической соли. Изменение pH также может быть эффективным для диссоциации иммунохимической связи между антигеном и аффинным матриксом.
[0407] "Средство" представляет собой низкомолекулярное химическое соединение, антитело, молекулу нуклеиновой кислоты или полипептид, или их фрагменты.
[0408] "Изменение" или "смена" представляет собой повышение или снижение. Изменение может составлять только 1%, 2%, 3%, 4%, 5%, 10%, 20%, 30% или 40%, 50%, 60% или даже 70%, 75%, 80%, 90% или 100%.
[0409] "Биологический образец" представляет собой любую ткань, клетку, жидкость или другой материал, происходящий из организма. Как используют в рамках изобретения, термин "образец" включает биологический образец, такой как любая ткань, клетка, жидкость или другой материал, происходящий из организма. "Специфически связывает" относится к соединению (например, пептид), которое распознает и связывает молекулу (например, полипептид), но по существу не распознает и не связывает другие молекулы в образце, например, в биологическом образце.
[0410] "Улавливающий реагент" относится к реагенту, который специфически связывает молекулу (например, молекула нуклеиновой кислоты или полипептид) для селекции или выделения молекулы (например, молекула нуклеиновой кислоты или полипептид).
[0411] Как используют в рамках изобретения, термины "определение", "оценка", "анализ", "измерение", "детекция" и их грамматические эквиваленты относятся как к количественному, так и к качественному определению и по существу термин "определение" используется в настоящем описании взаимозаменяемо с "анализом", "измерением" и т.п. Когда предполагается количественное определение, используют выражение "определение количества" анализируемого соединения и т.п. Когда подразумевается качественное и/или количественное определение, используют выражение "определение уровня" анализируемого соединения или "детекция" анализируемого соединения.
[0412] "Фрагмент" представляет собой часть белка или нуклеиновой кислоты, которая по существу идентична эталонному белку или нуклеиновой кислоте. В некоторых вариантах осуществления часть сохраняет по меньшей мере 50%, 75%, или 80%, или 90%, 95% или даже 99% биологической активности эталонного белка или нуклеиновой кислоты, описанных в настоящем описании.
[0413] Термины "выделенный", "очищенный", "биологически чистый" и их грамматические эквиваленты относятся к материалу, который свободен в различной степени от компонентов, которые обычно сопровождают его в нативном состоянии. "Выделять" означает степень отделения от оригинального источника или окружения. "Очищать" означает степень отделения, превышающую выделение. "Очищенный" или "биологически чистый" белок достаточно свободен от других материалов, так что какие-либо примеси не оказывают значительного влияния на биологические свойства белка или не вызывают других неблагоприятных последствий. Таким образом, нуклеиновая кислота или пептид по настоящему изобретению является очищенной, если она по существу свободна от клеточного материала, вирусного материала или культуральной среды, когда они продуцированы способами рекомбинантных ДНК, или химических предшественников или других химических реагентов, когда они химически синтезированы. Чистоту и гомогенность, как правило, определяют с использованием способов аналитической химии, например, полиакриламидного гель-электрофореза или высокоэффективной жидкостной хроматографии. Термин "очищенный" может означать, что нуклеиновая кислота или белок дает начало по существу одной полосе в электрофоретическом геле. Для белка, который может быть подвергнут модификациям, например, фосфорилированию или гликозилированию, различные модификации могут давать начало различным выделенным белкам, которые могут быть очищены по отдельности.
[0414] "Выделенный" полипептид (например, пептид из комплекса HLA-пептид) или полипептидный комплекс (например, комплекс HLA-пептид) представляет собой полипептид или полипептидный комплекс по настоящему изобретению, который отделен от компонентов, которые в природе сопровождают его. Как правило, полипептид или полипептидный комплекс является выделенным, когда он по меньшей мере на 60% по массе свободен от белков и встречающихся в природе органических молекул, с которыми он в природе ассоциирован. Препарат также может представлять собой по меньшей мере 75%, по меньшей мере 90%, или по меньшей мере 99% по массе полипептид или полипептидный комплекс по настоящему изобретению. Выделенный полипептид или полипептидный комплекс по настоящему изобретению может быть получен, например, посредством экстракции из природного источника, посредством экспрессии рекомбинантной нуклеиновой кислоты, кодирующей такой полипептид или один или более компонентов полипептидного комплекса, или посредством химического синтеза полипептида или одного или более компонентов полипептидного комплекса. Чистоту можно определять любым подходящим способом, например, колоночной хроматографией, полиакриламидным гель-электрофорезом или посредством ВЭЖХ-анализа. В некоторых случаях кодируемый аллелем HLA белок MHC класса II (т.е. пептид MHC класса II) взаимозаменяемо обозначается в настоящем описании как белок HLA класса II (или пептид HLA класса II).
[0415] Термин "векторы" относится к молекуле нуклеиновой кислоты, способной транспортировать или опосредовать экспрессию гетерологичной нуклеиновой кислоты. Плазмида представляет собой вид из рода, охватываемого термином "вектор". Вектор, как правило, относится к последовательности нуклеиновой кислоты, содержащей ориджин репликации и другие элементы, необходимые для репликации и/или поддержания в клетке-хозяине. Векторы, способные направлять экспрессию генов и/или последовательности нуклеиновой кислоты, с которой они функционально связаны, называют в настоящем описании "экспрессирующими векторами". Как правило, пригодные экспрессирующие векторы часто имеют форму "плазмид", которые относятся к кольцевым двухцепочечным молекулам ДНК, которые в их векторной форме не связаны с хромосомой, и, как правило, содержат элементы для стабильной или временной экспрессии или кодируемую ДНК. Другие экспрессирующие векторы, которые могут использоваться в способах, описанных в настоящем описании, включают, но не ограничиваются ими, плазмиды, эписомы, бактериальные искусственные хромосомы, дрожжевые искусственные хромосомы, бактериофаги или вирусные векторы, и такие векторы могут встраиваться в геном хозяина или автономно реплицироваться в клетке. Вектор может представлять собой ДНК- или РНК-вектор. Также можно использовать другие формы экспрессирующих векторов, известные специалистам в данной области, которые выполняют эквивалентные функции, например, самореплицирующиеся внехромосомные векторы или векторы, способные встраиваться в геном хозяина. Иллюстративные векторы представляют собой векторы, способные к автономной репликации и/или экспрессии нуклеиновых кислот, с которыми они связаны.
[0416] Термины "спейсер" или "линкер", как используют в отношении слитого белка, относятся к пептиду, который соединяет белки, составляющие слитый белок. Как правило, спейсер не имеет биологической активности, отличной от соединения или сохранения некоторого минимального расстояния или другой пространственной взаимосвязи между последовательностями белков или РНК. Однако в некоторых вариантах осуществления аминокислоты, составляющие спейсер, могут быть выбраны так, чтобы влиять на некоторое свойство молекулы, такое как укладка, суммарный заряд или гидрофобность молекулы. Подходящие линкеры для применения в одном из вариантов осуществления настоящего изобретения хорошо известны специалистам в данной области и включают, но не ограничиваются ими, прямые или разветвленные углеродные линкеры, гетероциклические углеродные линкеры или пептидные линкеры. Линкер используют для разделения двух антигенных пептидов на расстояние, достаточное, чтобы обеспечить, в некоторых вариантах осуществления, надлежащую укладку каждого антигенного пептида. Иллюстративные пептидные линкерные последовательности имеют гибкую протяженную конформацию и не демонстрируют склонности к формированию упорядоченной вторичной структуры. Типичные аминокислоты в подвижных областях белков включают Gly, Asn и Ser. Можно ожидать, что практически любой вариант перестановки аминокислотных последовательностей, содержащих Gly, Asn и Ser, будет удовлетворять описанным выше критериям для линкерной последовательности. В линкерной последовательности также могут использоваться другие практически нейтральные аминокислоты, такие как Thr и Ala. Другие аминокислотные последовательности, которые могут использоваться в качестве линкеров, описаны в Maratea et al. (1985), Gene 40: 39-46; Murphy et al. (1986) Proc. Nat'l. Acad. Sci. USA 83: 8258-62; патенте США №4935233; и патенте США №4751180.
[0417] Термин "новообразование" относится к любому заболеванию, которое вызывается или приводит к ненадлежаще высоким уровням деления клеток, ненадлежаще низким уровням апоптоза, или обоим из них. Неограничивающим примером новообразования или злокачественной опухоли является глиобластома. Термины "злокачественная опухоль", или "опухоль", или "гиперпролиферативное нарушение" относятся к присутствию клеток, обладающих характеристиками, типичными для вызывающих злокачественную опухоль клеток, такими как неконтролируемая пролиферация, бессмертие, потенциал к метастазированию, быстрый рост и пролиферация и определенные характерные морфологические признаки. Злокачественные клетки часто имеют форму опухоли, однако такие клетки могут существовать отдельно в животном или могут представлять собой не образующую опухоль злокачественную клетку, такую лейкозная клетка. Злокачественные клетки включают, но не ограничиваются ими, B-клеточную злокачественную опухоль (например, множественная миелома, макроглобулинемия Вальденстрема), болезни тяжелых цепей (например, такие как болезнь альфа-цепи, болезнь гамма-цепи и болезнь мю-цепи), доброкачественную моноклональную гаммапатию и иммуноцитарный амилоидоз, меланомы, рак молочной железы, рак легкого, рак бронхов, рак ободочной и прямой кишки, рак предстательной железы (например, метастазирующий, рефрактерный к гормонам рак предстательной железы), рак поджелудочной железы, рак желудка, рак яичника, рак мочевого пузыря, рак головного мозга или центральной нервной системы, рак периферической нервной системы, рак пищевода, рак шейки матки, рак матки или эндометрия, рак полости рта или глотки, рак печени, рак почки, рак яичка, рак желчных путей, рак тонкого кишечника или аппендикса, рак слюнных желез, рак щитовидной железы, рак надпочечника, остеосаркому, хондросаркому, рак гематологических тканей и т.п. Другие неограничивающие примеры типов злокачественных опухолей, для которых применимы способы, охватываемые настоящим изобретением, включают саркомы и карциномы человека, например, фибросаркому, миксосаркому, липосаркому, хондросаркому, остеогенную саркому, хордому, ангиосаркому, эндотелиосаркому, лимфангиосаркому, лимфангиоэндотелиосаркому, синовиому, мезотелиому, опухоль Юинга, лейомиосаркому, рабдомиосаркому, карциному толстого кишечника, рак ободочной и прямой кишки, рак поджелудочной железы, рак молочной железы, рак яичника, плоскоклеточную карциному, базально-клеточную карциному, аденокарциному, карциному потовых желез, карциному сальных желез, папиллярную карциному, папиллярную аденокарциному, цистаденокарциному, медуллярную карциному, бронхогенную карциному, почечноклеточный рак, гепатому, карциному желчных протоков, рак печени, хориокарциному, семиному, эмбриональную карциному, опухоль Вильмса, рак шейки матки, рак кости, опухоль головного мозга, рак яичка, карциному легкого, мелкоклеточную карциному легкого, карциному мочевого пузыря, эпителиальную карциному, глиому, астроцитому, медуллобластому, краниофарингиому, эпендимоме, пинеалому, геманиобластому, невриному слухового нерва, олигоденроглиому, менингиому, меланому, нейробластому, ретинобластому; лейкозы, например, острый лимфоцитарный лейкоз и острый миелоцитарный лейкоз (миелобластный, промиелоцитарный, миеломоноцитарный, моноцитарный и эритролейкоз); хронический лейкоз (хронический миелоцитарный (гранулоцитарный) лейкоз и хронический лимфоцитарный лейкоз); и истинную полицитемию, лимфому (болезнь Ходжкина и неходжкинскую болезнь), множественную миелому, макроглобулинемию вальденстрема и болезнь тяжелых цепей. В некоторых вариантах осуществления злокачественная опухоль представляет собой эпителиальную злокачественную опухоль, такую как, но не ограничиваясь ими, рак мочевого пузыря, рак молочной железы, рак шейки матки, рак толстого кишечника, гинекологические злокачественные опухоли, рак почки, рак гортани, рак легкого, рак полости рта, рак головы и шеи, рак яичника, рак поджелудочной железы, рак предстательной железы или рак кожи. В других вариантах осуществления злокачественная опухоль представляет собой рак молочной железы, рак предстательной железы, рак легкого или рак толстого кишечника. В других вариантах осуществления эпителиальный рак представляет собой немелкоклеточный рак легкого, непапиллярный почечноклеточный рак, карциному шейки матку, карциному яичника (например, серозная карцинома яичника) или карциному молочной железы. Эпителиальные злокачественные опухоли могут быть охарактеризованы по-разному, включая, но не ограничиваясь ими, серозные, эндометриоидные, муцинозные, светлоклеточные, опухоли Бреннера или недифференцированные. В некоторых вариантах осуществления настоящее изобретение используется для лечения, диагностики и/или прогнозирования лимфомы или ее подтипов, включая, но не ограничиваясь ими, лимфому из клеток мантийной зоны. Лимфопролиферативные нарушения также считаются пролиферативными заболеваниями.
[0418] Под термином "вакцина" понимают композицию для индукции иммунитета для профилактики и/или лечения заболеваний (например, новообразование/опухоль/инфекционные агенты/аутоиммунные заболевания). Таким образом, вакцины представляют собой лекарственные средства, которые содержат антигены и предназначены для применения у человека или животных для индукции специфической защиты и протективных веществ посредством вакцинации. "Вакцинная композиция" может включать фармацевтически приемлемый эксципиент, носитель или разбавитель. Аспекты настоящего изобретения относятся к применению технологии получения вакцины на основе антигенов. В этих вариантах осуществления под вакциной понимают один или более специфических для заболевания антигенных пептидов (или соответствующих нуклеиновых кислот, кодирующих их). В некоторых вариантах осуществления вакцина на основе антигена содержит по меньшей мере два, по меньшей мере три, по меньшей мере четыре, по меньшей мере пять, по меньшей мере шесть, по меньшей мере семь, по меньшей мере восемь, по меньшей мере девять, по меньшей мере 10, по меньшей мере 11, по меньшей мере 12, по меньшей мере 13,по меньшей мере 14, по меньшей мере 15, по меньшей мере 16, по меньшей мере 17, по меньшей мере 18, по меньшей мере 19, по меньшей мере 20, по меньшей мере 21, по меньшей мере 22, по меньшей мере 23, по меньшей мере 24, по меньшей мере 25, по меньшей мере 26, по меньшей мере 27, по меньшей мере 28, по меньшей мере 29, по меньшей мере 30 или более антигенных пептидов. В некоторых вариантах осуществления вакцина на основе антигена содержит от 2 до 100, от 2 до 75, от 2 до 50, от 2 до 25, от 2 до 20, от 2 до 19, от 2 до 18, от 2 до 17, от 2 до 16, от 2 до 15, от 2 до 14, от 2 до 13, от 2 до 12, от 2 до 10, от 2 до 9, от 2 до 8, от 2 до 7, от 2 до 6, от 2 до 5, от 2 до 4, от 3 до 100, от 3 до 75, от 3 до 50, от 3 до 25, от 3 до 20, от 3 до 19, от 3 до 18, от 3 до 17, от 3 до 16, от 3 до 15, от 3 до 14, от 3 до 13, от 3 до 12, от 3 до 10, от 3 до 9, от 3 до 8, от 3 до 7, от 3 до 6, от 3 до 5, от 4 до 100, от 4 до 75, от 4 до 50, от 4 до 25, от 4 до 20, от 4 до 19, от 4 до 18, от 4 до 17, от 4 до 16, от 4 до 15, от 4 до 14, от 4 до 13, от 4 до 12, от 4 до 10, от 4 до 9, от 4 до 8, от 4 до 7, от 4 до 6, от 5 до 100, от 5 до 75, от 5 до 50, от 5 до 25, от 5 до 20, от 5 до 19, от 5 до 18, от 5 до 17, от 5 до 16, от 5 до 15, от 5 до 14, от 5 до 13, от 5 до 12, от 5 до 10, от 5 до 9, от 5 до 8, или от 5 до 7 антигенных пептидов. В некоторых вариантах осуществления вакцина на основе антигена содержит 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19 или 20 антигенных пептидов. В некоторых случаях антигенные пептиды представляют собой неоантигенные пептиды. В некоторых случаях антигенные пептиды содержат один или более неоэпитопов.
[0419] Термин "фармацевтически приемлемый" относится к одобренному или заслуживающему одобрения регуляторным органом федерального правительства или правительства штата, или приведенный в фармакопее США или другой общепризнанной фармакопее для применения у животных, в том числе человека. "Фармацевтически приемлемый эксципиент, носитель или разбавитель" относится к эксципиенту, носителю или разбавителю, который можно вводить индивидууму вместе со средством и который не нарушает его фармакологическую активность и является нетоксичным при введении в дозах, достаточных для доставки терапевтического количества средства. "Фармацевтически приемлемая соль" для специфических для заболевания антигенов в совокупности, как упоминается в настоящем описании, может представлять собой кислотную или основную соль, которая, как правило, считается в данной области пригодной для применения в контакте с тканями человека или животных без чрезмерной токсичности, раздражения, аллергического ответа или другой проблемы или осложнения. Такие соли включают минеральные и органические кислотные соли основных остатков, таких как амины, а также щелочные или органические соли кислотных остатков, таких как карбоновые кислоты. Конкретные фармацевтические соли включают, но не ограничиваются ими, соли кислот, таких как хлористоводородная, фосфорная, бромистоводородная, яблочная, гликолевая, фумаровая, серная, сульфамовая, сульфаниловая, муравьиная, толуолсульфоновая, метансульфоновая, бензолсульфоновая, этандисульфоновая, 2-гидроксиэтилсульфоновая, азотная, бензойная, 2-ацетоксибензойная, лимонная, виннокаменная, молочная, стеариновая, салициловая, глутаминовая, аскорбиновая, памовая, янтарная, фумаровая, малеиновая, пропионовая, гидроксималеиновая, йодистоводородная, фенилуксусная, алкановая, такая как уксусная, HOOC-(CH2)n-COOH, где n равен 0-4, и т.п. Аналогично, фармацевтически приемлемые катионы включают, но не ограничиваются ими, натрий, калий, кальций, алюминий, литий и аммоний. Специалистам в данной области из настоящего описания и информации в данной области известны другие фармацевтически приемлемые соли специфических для заболевания антигенов в совокупности, описанных в настоящем описании, включая соли, приведенные в Remington's Pharmaceutical Sciences, 17th ed., Mack Publishing Company, Easton, PA, p. 1418 (1985). Как правило, фармацевтически приемлемую кислотную или основную соль можно синтезировать из исходного соединения, которое содержит основную или кислотную часть, любым общепринятым химическим способом. В кратком изложении, такие соли можно получать путем реакции форм свободной кислоты или свободного основания этих соединений со стехиометрическим количеством соответствующего основания или кислоты в подходящем растворителе.
[0420] Молекулы нуклеиновых кислот, пригодные в способах по изобретению, включают любую молекулу нуклеиновой кислоты, которая кодирует полипептид по изобретению или его фрагмент. Такая молекула нуклеиновой кислоты не должна быть на 100% идентичной эндогенной последовательности нуклеиновой кислоты, но, как правило, демонстрирует существенную идентичность. Полинуклеотиды, обладающие существенной идентичностью с эндогенной последовательностью, как правило, способны гибридизоваться по меньшей мере с одной цепью двухцепочечной молекулы нуклеиновой кислоты. "Гибридизоваться" относится к тому, когда молекулы нуклеиновых кислот образуют пару с образованием двухцепочечной молекулы между комплементарными полинуклеотидными последовательностями или их частями в условиях различной жесткости. (См., например, Wahl, G.M. and S.L. Berger (1987) Methods Enzymol. 152:399; Kimmel, A.R. (1987) Methods Enzymol. 152:507). Например, жесткие условия концентрации соли обычно могут составлять менее чем приблизительно 750 мМ NaCl и 75 мМ трицитрат натрия, менее чем приблизительно 500 мМ NaCl и 50 мМ трицитрат натрия, или менее чем приблизительно 250 мМ NaCl и 25 мМ трицитрат натрия. Гибридизации низкой жесткости можно достигать в отсутствии органического растворителя, например, формамида, в то время как гибридизации высокой жесткости можно достигать в присутствии по меньшей мере приблизительно 35% формамида или по меньшей мере приблизительно 50% формамида. Жесткие условия температуры обычно могут включать температуру по меньшей мере приблизительно 30°C, по меньшей мере приблизительно 37°C или по меньшей мере приблизительно 42°C. Различные дополнительные параметры, такие как время гибридизации, концентрация детергента, например, додецилсульфата натрия (SDS), и включение или исключение ДНК-переносчика, известны специалистам в данной области. Различных уровней жесткости достигают путем комбинирования этих различных условий по необходимости. В иллюстративных вариантах осуществления гибридизация может происходить при 30° в 750 мМ NaCl, 75 мМ трицитрате натрия и 1% SDS. В другом иллюстративном варианте осуществления гибридизация может происходить при 37°C в 500 мМ NaCl, 50 мМ трицитрате натрия, 1% SDS, 35% формамиде и 100 мкг/мл денатурированной ДНК спермы лосося (ss-ДНК). В другом иллюстративном варианте осуществления гибридизация может происходить при 42°C в 250 мМ NaCl, 25 мМ трицитрате натрия, 1% SDS, 50% формамиде и 200 мкг/мл ss-ДНК. Подходящие изменения этих условий будут хорошо понятны специалистам в данной области. Для большинства применений стадии промывания после гибридизации также могут варьироваться в отношении жесткости. Условия жесткости промывания могут определяться концентрацией соли и температурой. Как описано выше, жесткость промывания может увеличиваться путем снижения концентрации соли или повышения температуры. Например, жесткая концентрация соли для стадий промывания может представлять собой менее чем приблизительно 30 мМ NaCl и 3 мМ трицитрат натрия, или менее чем приблизительно 15 мМ NaCl и 1,5 мМ трицитрат натрия. Жесткие температурные условия для стадий промывания могут включать температуру по меньшей мере приблизительно 25°C, по меньшей мере приблизительно 42°C или по меньшей мере приблизительно 68°C. В иллюстративных вариантах осуществления стадии промывания могут происходить при 25°C в 30 мМ NaCl, 3 мМ трицитрате натрия и 0,1% SDS. В других иллюстративных вариантах осуществления стадии промывания могут происходить при 42°C в 15 мМ NaCl, 1,5 мМ трицитрате натрия и 0,1% SDS. В другом иллюстративном варианте осуществления стадии промывания могут происходить при 68°C в 15 мМ NaC1, 1,5 мМ трицитрате натрия и 0,1% SDS. Дополнительное варьирование этих условий может быть хорошо понятно специалистам в данной области. Способы гибридизации хорошо известны специалистам в данной области и описаны, например, в Benton and Davis (Science 196:180, 1977); Grunstein and Hogness (Proc. Natl. Acad. Sci., USA 72:3961, 1975); Ausubel et al. (Current Protocols in Molecular Biology, Wiley Interscience, New York, 2001); Berger and Kimmel (Guide to Molecular Cloning Techniques, 1987, Academic Press, New York); и Sambrook et al., Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory Press, New York.
[0421] "По существу идентичный" относится к полипептиду или молекуле нуклеиновой кислоты, демонстрирующим по меньшей мере 50% идентичность с эталонной аминокислотной последовательностью (например, любой из аминокислотных последовательностей, описанных в настоящем описании) или последовательностью нуклеиновой кислоты (например, любая из последовательностей нуклеиновых кислот, описанных в настоящем описании). Такая последовательность может быть по меньшей мере на 60%, 80% или 85%, 90%, 95%, 96%, 97%, 98%, или даже 99% или более идентичной на уровне аминокислот или нуклеиновой кислоты с последовательностью, используемой для сравнения. Идентичность последовательностей, как правило, определяют с использованием программного обеспечения для анализа последовательностей (например, Sequence Analysis Software Package of the Genetics Computer Group, University of Wisconsin Biotechnology Center, 1710 University Avenue, Madison, Wis. 53705, программы BLAST, BESTFIT, GAP или PILEUP/PRETTYBOX). Такое программное обеспечение сопоставляет идентичные или сходные последовательности путем присвоения степеней гомологии различным заменам, делециям и/или другим модификациям. Консервативные замены, как правило, включают замены из следующих групп: глицин, аланин; валин, изолейцин, лейцин; аспарагиновая кислота, глутаминовая кислота, аспарагин, глутамин; серин, треонин; лизин, аргинин и фенилаланин, тирозин. В иллюстративном подходе для определения степени идентичности можно использовать программу BLAST с показателем вероятности между e-3 и e-m°, указывающим на близкородственную последовательность. "Эталон" представляет собой стандарт для сравнения.
[0422] Термин "индивидуум" или "пациент" относится к животному, которое является объектом лечения, наблюдения или эксперимента. Только в качестве примера, индивидуум включает, но не ограничивается ими, млекопитающее, включая, но не ограничиваясь ими, человека или не являющееся человеком млекопитающее, такое как не являющийся человеком примат, животное семейства мышиных, животное подсемейства бычьих, животное семейства лошадиных, животное семейства собачьих, животное семейства овечьих или животное семейства кошачьих.
[0423] Подразумевается, что термины "лечить", "подвергнутый лечению", "лечащий", "лечение" и т.п. относятся к снижению, предупреждению или смягчению нарушения и/или симптомов, ассоциированных с ним (например, новообразование, или опухоль, или инфекционный агент, или аутоиммунное заболевание). "Лечение" может относиться к проведению терапии у индивидуума после начала или предполагаемого начала заболевания (например, злокачественная опухоль, или инфекция инфекционным агентом, или аутоиммунное заболевание). "Лечение" включает концепцию "облегчения", которое относится к уменьшению частоты возникновения или рецидива, или тяжести любых симптомов или других эффектов заболевания, связанных с заболеванием, и/или побочных эффектов, ассоциированных с терапией. Термин "лечение" также охватывает концепцию "управления течением", которое относиться к снижению тяжести заболевания или нарушения у пациента, например, увеличению продолжительности жизни или продлению выживания пациента с заболеванием, или отсрочиванию его рецидива, например, увеличению периода ремиссии у пациента, который страдал от заболевания. Понятно, что, хотя и не исключительно, лечение нарушения или состояния не требует, чтобы нарушение, состояние или симптомы, ассоциированные с ним, полностью устранялись.
[0424] Термин "предупреждать", "предупреждающий", "предупреждение" и их грамматические эквиваленты, как используют в рамках изобретения, означает избегание или отсрочивание появления симптомов, ассоциированных с заболеванием или состоянием у индивидуума, у которого не развились такие симптомы на момент начала введения средства или соединения.
[0425] Термин "терапевтический эффект" относится к некоторой степени облегчения одного или более симптомов нарушения (например, новообразование, опухоль или инфекция посредством инфекционного агента или аутоиммунное заболевание) или ассоциированной с ним патологии. "Терапевтически эффективное количество", как используют в рамках изобретения, относится к количеству средства, которое является эффективным при введении однократной или многократной дозы в клетку или индивидууму для продления выживания пациента с таким нарушением, уменьшения одного или более признаков или симптомов нарушения, предупреждения их или отсрочивание и т.п. за пределы ожидаемого в отсутствие такого лечения. Подразумевают, что "терапевтически эффективное количество" определяет количество, требуемое для достижения терапевтического эффекта. Врач или ветеринар, являющийся средним специалистом в данной области, может без труда определить и назначить "терапевтически эффективное количество" (например, ED50) требуемой фармацевтической композиции. Например, врач или ветеринар может начать дозирование соединений по настоящему изобретению, используемых в фармацевтической композиции, на более низких уровнях, чем требуется для достижения желаемого терапевтического эффекта, и постепенно повышать дозировку до тех пор, пока не будет достигнут желаемый эффект. Заболевание, состояние и нарушение используют в настоящем описании взаимозаменяемо.
[0426] Специалистам в данной области будет понятно, что термины "пептидная метка", "аффинная метка", "эпитопная метка" или "аффинная акцепторная метка" используют в настоящем описании взаимозаменяемо. Как используют в рамках изобретения, термин "аффинная акцепторная метка" относится к аминокислотной последовательности, которая позволяет без труда выявить или очистить меченый белок, например, посредством аффинной очистки. Аффинную акцепторную метку, как правило (но не обязательно), помещают на или вблизи N- или C-конца аллеля HLA. Различные пептидные метки хорошо известны в данной области. Неограничивающие примеры включают полигистидиновую метку (например, 4-15 последовательно расположенных остатков His (SEQ ID NO: 4), как например, 8 последовательно расположенных остатков His (SEQ ID NO: 5)); поли-гистидин-глициновую метку; HA-метку (например, Field et al., Mol. Cell. Biol., 8:2159, 1988); c-myc-метку (например, Evans et al., Mol. Cell. Biol., 5:3610, 1985); метку гликопротеина D (gD) вируса простого герпеса (например, Paborsky et al., Protein Engineering, 3:547, 1990); FLAG-метку (например, Hopp et al., BioTechnology, 6:1204, 1988; патенты США №4703004 и 4851341); эпитопную метку KT3 (например, Martine et al., Science, 255:192, 1992); эпитопную метку тубулина (например, Skinner, Biol. Chem., 266:15173, 1991); пептидную метку белка гена 10 T7 (например, Lutz-Freyemuth et al., Proc. Natl. Acad. Sci. USA, 87:6393, 1990); стрептавидиновую метку (StrepTag.TM. или StrepTagII.TM.; см., например, Schmidt et al., J. Mol. Biol., 255(5):753-766, 1996 или патент США №5506121; также коммерчески доступные от Sigma-Genosys); или эпитопную метку VSV-G, происходящую из гликопротеина вируса везикулярного стоматита; или V5-метку, происходящую из небольшого эпитопа (Pk), находящегося на белках P и V парамиксовируса, вируса обезьян 5 (SV5). В некоторых вариантах осуществления аффинная акцепторная метка представляет собой "эпитопную метку", которая представляет собой тип пептидной метки, которая добавляет распознаваемый эпитоп (участок связывания антитела) к HLA-белку для обеспечения связывания соответствующего антитела, тем самым позволяя идентификацию или аффинную очистку меченого белка. Неограничивающий пример эпитопной метки представляет собой белок A или белок G, который связывается с IgG. В некоторых вариантах осуществления матрикс хроматографической смолы IgG Sepharose 6 Fast Flow ковалентно связан с IgG человека. Эта смола позволяет высокие скорости потока, быструю и удобную очистку белка, меченного белком A. Многочисленные другие части меток известны и могут быть предусмотрены специалистом в данной области, и они охватываются настоящим изобретением. Можно использовать любую пептидную метку при условии, что она может экспрессироваться в качестве элемента комплекса HLA-пептид, меченного аффинной акцепторной меткой.
[0427] Как используют в рамках изобретения, термин "аффинная молекула" относится к молекуле или лиганду, которая связывается с химической специфичностью с аффинным акцепторным пептидом. Химическая специфичность представляет собой способность связывающего участка в белке связывать определенные лиганды. Чем меньше лигандов может связывать белок, тем больше его специфичность. Специфичность описывает силу связывания между данным белком и лигандом. Эта взаимосвязь может быть описана посредством константы диссоциации (KD), которая характеризует равновесие между связанным и несвязанным состояниями системы белок-лиганд.
[0428] Термин "комплекс HLA-пептид, меченный аффинной акцепторной меткой" относится к комплексу, содержащему пептид, связывающийся с HLA класса I или класса II, или его часть, специфически связанный с одноаллельным рекомбинантным пептидом HLA класса I или класса II, содержащим аффинный акцепторный пептид.
[0429] Термины "специфическое связывание" или "специфически связывающийся", когда их используют в отношении взаимодействия аффинной молекулы и аффинной акцепторной метки или эпитопа и пептида HLA, означает, что взаимодействие зависит от присутствия конкретной структуры (например, антигенной детерминанты или эпитопа) на белке; иными словами аффинная молекула распознает и связывает определенную структуру аффинного акцепторного пептида, а не белки в целом.
[0430] Как используют в рамках изобретения термин "аффинность" относится к показателю силы связывания между двумя представителями связывающейся пары, например, "аффинной акцепторной метки" и "аффинной молекулой", и HLA-связывающим пептидом и молекулой HLA класса I или II. KD представляет собой константу диссоциации, и она имеет единицы молярности. Константа аффинности представляет собой обратную величину константы диссоциации. Константу аффинности иногда используют в качестве общего термина для описания данной химической структуры. Она является прямым показателем энергии связывания. Аффинность можно определять экспериментально, например, посредством поверхностного плазмонного резонанса (SPR), с использованием коммерчески доступных элементов SPR Biacore. Аффинность также может быть выражена в качестве ингибиторной концентрации 50 (IC50), такой концентрации, при которой вытесняется 50% пептида. Аналогично, lnIC50 относится к натуральному логарифму IC50. Koff относится к константе скорости диссоциации, например, для диссоциации аффинной молекулы из комплекса HLA-пептид, меченного аффинной акцепторной меткой.
[0431] В некоторых вариантах осуществления комплекс HLA-пептид, меченный аффинной акцепторной меткой, включает биотин-акцепторный пептид (BAP), и его подвергают иммунной очистке из комплексных клеточных смесей с использованием гранул стрептавидин/нейтравидин. Связывание биотин-авидин/стрептавидин является наиболее сильным нековалентным взаимодействием, известным в природе. Это свойство используется в качестве биологического инструмента для широкого диапазона применений, таких как иммунная очистка белка, с которым биотин ковалентно связан. В иллюстративном варианте осуществления последовательность нуклеиновой кислоты, кодирующая аллель HLA, включает биотин-акцепторный пептид (BAP) в качестве аффинной акцепторной метки для иммунной очистки. BAP может быть специфически биотинилирован in vivo или in vitro на одном остатке лизина в метке (например, патенты США №5723584; 5874239; и 5932433; и патент Великобритании № GB2370039). BAP, как правило, имеет длину 15 аминокислот и содержит один остаток лизина в качестве акцепторного остатка для биотина. В некоторых вариантах осуществления BAP находится на или вблизи N- или C-конца одноаллельного пептида HLA. В некоторых вариантах осуществления BAP находится между доменом тяжелой цепи и доменом β2-микроглобулина в пептиде HLA класса I. В некоторых вариантах осуществления BAP находится между доменом β-цепи и доменом α-цепи пептида HLA класса II. В некоторых вариантах осуществления BAP находится в петлевых областях между доменами α1, α2 и α3 тяжелой цепи HLA класса I, или между доменами α1 и α2 и β1 и β2 α-цепи и β-цепи, соответственно HLA класса II. Иллюстративные конструкции, сконструированные для экспрессии HLA класса I и II с использованием BAP для биотинилирования и иммунной очистки, описаны на фиг. 2.
[0432] Как используют в рамках изобретения, термин "биотин" относится к самому соединению биотина и его аналогам, производным и вариантам. Таким образом, термин "биотин" включает биотин (цис-гексагидро-2-оксо-1H-тиено[3,4]имидазол-4-пентановая кислота) и любые его производные и аналоги, включая биотин-подобные соединения. Такие соединения включают, например, биотин-e-N-лизин, биоцитин гидразид, амино или сульфгидрильные производные 2-иминобиотина и N-гидроксисукцинимидный сложный эфир биотинил-E-аминокапроновой кислоты, сульфосукцинимидиминобиотин, биотинбромацетилгидразид, п-диазобензоилбиоцитин, 3-(N-малеимидопропионил)биоцитин, дестиобиотин и т.п. Термин "биотин" также включает варианты биотина, которые могут специфически связываться с одной или более из частей ризавидина, авидина, стрептавидина, тамавидина, или другими авидин-подобными пептидами.
[0433] Как используют в рамках изобретения, "способ определения PPV" может относиться к способу определения PPV презентации. Например, "способ определения PPV" может относиться к способу, включающему (a) обработку информации об аминокислотах для множества тестовых пептидных последовательностей с использованием модели прогнозирования презентации пептидов посредством HLA, такой как машинно-обучаемая модель для прогнозирования презентации пептидов HLA, с получением множества тестовых прогнозов презентации, причем каждый тестовый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II в клетке, таким как аллель HLA класса II в клетке индивидуума, может презентировать данную тестовую пептидную последовательность из множества тестовых пептидных последовательностей, где множество тестовых пептидных последовательностей включает по меньшей мере 500 тестовых пептидных последовательностей, содержащих (i) по меньшей мере одну наиболее подходящую пептидную последовательность, идентифицированную посредством масс-спектрометрии в качестве презентируемой белком HLA, экспрессируемым в клетках и (ii) по меньшей мере 499 последовательностей пептидов-ловушек, содержащихся в белке, кодируемом геномом организма, таком как организм, принадлежащий к тому же виду, что и индивидуум, где множество тестовых пептидных последовательностей включает соотношение, составляющее менее единицы, количества наиболее подходящих пептидных последовательностей и количества пептидов-ловушек, такое как соотношение 1:499 для по меньшей мере одной из наиболее подходящих пептидных последовательностей и по меньшей мере 499 последовательностей пептидов-ловушек; (b) идентификацию или назначение наивысшего процента из множества тестовых последовательностей, такого как наивысшие 0,2% из множества тестовых пептидных последовательностей, в качестве презентируемых аллелем HLA класса II клетки; и (c) вычисление PPV для модели прогнозирования презентации пептидов посредством HLA, где PPV представляет собой долю тестовых пептидных последовательностей из множества, которые идентифицированы или назначены как презентируемые аллелем HLA класса II клетки, которые представляют собой пептиды, выявленные посредством масс-спектрометрии в качестве презентируемых аллелем HLA класса II клетки. В некоторых вариантах осуществления пептид-ловушка имеет ту же длину, т.е. содержит такое же количество аминокислот, что и наиболее подходящий пептид. В некоторых вариантах осуществления пептид-ловушка может содержать на одну аминокислоту больше или на одну аминокислоту меньше по сравнению с наиболее подходящим пептидом. В некоторых вариантах осуществления пептид-ловушка представляет собой пептид, который представляет собой эндогенный пептид. В некоторых вариантах осуществления пептид-ловушка представляет собой синтетический пептид. В некоторых вариантах осуществления пептид-ловушка представляет собой эндогенный пептид, который идентифицирован посредством масс-спектрометрии в качестве связывающегося с первым белком MHC класса I или класса II, где первый белок MHC класса I или класса II отличается от второго белка MHC класса I или класса II, который связывается с наиболее подходящим пептидом. В некоторых вариантах осуществления пептид-ловушка может представлять собой перемешанный пептид, например, пептид-ловушка может включать аминокислотную последовательность, в которой положения аминокислот реорганизованы относительно положений аминокислот в наиболее подходящем пептиде в рамках длины пептида. В некоторых вариантах осуществления способ определения PPV может представлять собой способ определения презентации PPV. В некоторых вариантах осуществления соотношение количества наиболее подходящих пептидных последовательностей и количества последовательностей пептидов-ловушек составляет приблизительно 1:10, 1:20, 1:50, 1:100, 1:250, 1:500, 1:1000, 1:1500, 1:2000, 1:2500, 1:5000, 1:7500, 1:10000, 1:25000, 1:50000 или 1:100000. В некоторых вариантах осуществления по меньшей мере одна наиболее подходящая пептидная последовательность содержит по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 или 100 наиболее подходящих пептидных последовательностей. В некоторых вариантах осуществления по меньшей мере 499 последовательностей пептидов-ловушек включают по меньшей мере 500 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3200, 3300, 3400, 3500, 3600, 3700, 3800, 3900, 4000, 4100, 4200, 4300, 4400, 4500, 4600, 4700, 4800, 4900, 5000, 5100, 5200, 5300, 5400, 5500, 5600, 5700, 5800, 5900, 6000, 6100, 6200, 6300, 6400, 6500, 6600, 6700, 6800, 6900, 7000, 7100, 7200, 7300, 7400, 7500, 7600, 7700, 7800, 7900, 8000, 8100, 8200, 8300, 8400, 8500, 8600, 8700, 8800, 8900, 9000, 9100, 9200, 9300, 9400, 9500, 9600, 9700, 9800, 9900, 10000, 11000, 12000, 13000, 14000, 15000, 16000, 17000, 18000, 19000, 20000, 21000, 22000, 23000, 24000, 25000, 26000, 27000, 28000, 29000, 30000, 31000, 32000, 33000, 34000, 35000, 36000, 37000, 38000, 39000, 40000, 41000, 42000, 43000, 44000, 45000, 46000, 47000, 48000, 49000, 50000, 52500, 55000, 57500, 60000, 62500, 65000, 67500, 70000, 72500, 75000, 77500, 80000, 82500, 85000, 87500, 90000, 92500, 95000, 97500, 100000, 125000, 150000, 175000, 200000, 225000, 250000, 275000, 300000, 325000, 350000, 375000, 400000, 425000, 450000, 475000, 500000, 600000, 700000, 800000, 900000 или 1000000 последовательностей пептидов-ловушек. В некоторых вариантах осуществления по меньшей мере 500 тестовых пептидных последовательностей включают по меньшей мере 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3200, 3300, 3400, 3500, 3600, 3700, 3800, 3900, 4000, 4100, 4200, 4300, 4400, 4500, 4600, 4700, 4800, 4900, 5000, 5100, 5200, 5300, 5400, 5500, 5600, 5700, 5800, 5900, 6000, 6100, 6200, 6300, 6400, 6500, 6600, 6700, 6800, 6900, 7000, 7100, 7200, 7300, 7400, 7500, 7600, 7700, 7800, 7900, 8000, 8100, 8200, 8300, 8400, 8500, 8600, 8700, 8800, 8900, 9000, 9100, 9200, 9300, 9400, 9500, 9600, 9700, 9800, 9900, 10000, 11000, 12000, 13000, 14000, 15000, 16000, 17000, 18000, 19000, 20000, 21000, 22000, 23000, 24000, 25000, 26000, 27000, 28000, 29000, 30000, 31000, 32000, 33000, 34000, 35000, 36000, 37000, 38000, 39000, 40000, 41000, 42000, 43000, 44000, 45000, 46000, 47000, 48000, 49000, 50000, 52500, 55000, 57500, 60000, 62500, 65000, 67500, 70000, 72500, 75000, 77500, 80000, 82500, 85000, 87500, 90000, 92500, 95000, 97500, 100000, 125000, 150000, 175000, 200000, 225000, 250000, 275000, 300000, 325000, 350000, 375000, 400000, 425000, 450000, 475000, 500000, 600000, 700000, 800000, 900000 или 1000000 тестовых пептидных последовательностей. В некоторых вариантах осуществления идентификация или назначение наивысшего процента из множества тестовых пептидных последовательностей в качестве презентируемых аллелем HLA класса II клетки включает идентификацию или назначение наивысших 0,20%, 0,30%, 0,40%, 0,50%, 0,60%, 0,70%, 0,80%, 0,90%, 1,00%, 1,10%, 1,20%, 1,30%, 1,40%, 1,50%, 1,60%, 1,70%, 1,80%, 1,90%, 2,00%, 2,10%, 2,20%, 2,30%, 2,40%, 2,50%, 2,60%, 2,70%, 2,80%, 2,90%, 3,00%, 3,10%, 3,20%, 3,30%, 3,40%, 3,50%, 3,60%, 3,70%, 3,80%, 3,90%, 4,00%, 4,10%, 4,20%, 4,30%, 4,40%, 4,50%, 4,60%, 4,70%, 4,80%, 4,90%, 5,00%, 5,10%, 5,20%, 5,30%, 5,40%, 5,50%, 5,60%, 5,70%, 5,80%, 5,90%, 6,00%, 6,10%, 6,20%, 6,30%, 6,40%, 6,50%, 6,60%, 6,70%, 6,80%, 6,90%, 7,00%, 7,10%, 7,20%, 7,30%, 7,40%, 7,50%, 7,60%, 7,70%, 7,80%, 7,90%, 8,00%, 8,10%, 8,20%, 8,30%, 8,40%, 8,50%, 8,60%, 8,70%, 8,80%, 8,90%, 9,00%, 9,10%, 9,20%, 9,30%, 9,40%, 9,50%, 9,60%, 9,70%, 9,80%, 9,90%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20% в качестве презентируемых аллелем HLA класса II клетки. В некоторых вариантах осуществления клетка представляет собой моноаллельную клетку.
[0434] Как используют в рамках изобретения, "способ определения PPV" может относиться к способу определения PPV связывания. Например, "способ определения PPV" может относиться к способу, включающему (a) обработку информации об аминокислотах для множества тестовых пептидных последовательностей с использованием модели прогнозирования связывания пептидов посредством HLA, такой как машинно-обучаемая модель для прогнозирования связывания пептидов HLA, с получением множества тестовых прогнозов связывания, причем каждый тестовый прогноз связывания указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II клетки, таким как аллель HLA класса II клетки индивидуума, связывается с данной тестовой пептидной последовательностью из множества тестовых пептидных последовательностей, где множество тестовых пептидных последовательностей содержит по меньшей мере 20 тестовых пептидных последовательностей, содержащих (i) по меньшей мере одну наиболее подходящую пептидную последовательность, идентифицированную посредством масс-спектрометрии в качестве презентируемой белком HLA, экспрессируемым в клетках, и (ii) по меньшей мере 19 последовательностей пептидов-ловушек, содержащихся в белке, содержащем по меньшей мере одну пептидную последовательность, идентифицированную посредством масс-спектрометрии в качестве презентируемой белком HLA, экспрессируемым в клетках, где множество тестовых пептидных последовательностей включает соотношение, составляющее менее единицы, количества наиболее подходящих пептидных последовательностей и количества последовательностей пептидов-ловушек, такое как соотношение 1:19 для по меньшей мере одной из наиболее подходящих пептидных последовательностей и по меньшей мере 19 последовательностей пептидов-ловушек; (b) идентификацию или назначение наивысшего процента из множества тестовых последовательностей, такого как наивысше 5% из множества тестовых пептидных последовательностей, в качестве связывающихся с белком HLA; и (c) вычисление PPV для модели прогнозирования связывания HLA с пептидом, где PPV представляет собой долю тестовых пептидных последовательностей из множества, которые были идентифицированы или назначены в качестве связывающихся с аллелем HLA класса II клетке, которые представляют собой пептиды, выявленные посредством масс-спектрометрии в качестве презентируемых аллелем HLA класса II клетки. В некоторых вариантах осуществления соотношение количества наиболее подходящих пептидных последовательностей и количества последовательностей пептидов-ловушек составляет приблизительно 1:2, 1:3, 1:4, 1:5, 1:10, 1:20, 1:25, 1:30, 1:40, 1:50, 1:75, 1:100, 1:200, 1:250, 1:500 или 1:1000. В некоторых вариантах осуществления по меньшей мере одна наиболее подходящая пептидная последовательность содержит по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 или 100 наиболее подходящих пептидных последовательностей. В некоторых вариантах осуществления по меньшей мере 19 последовательностей пептидов-ловушек включают по меньшей мере 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200, 210, 220, 230, 240, 250, 260, 270, 280, 290, 300, 310, 320, 330, 340, 350, 360, 370, 380, 390, 400, 410, 420, 430, 440, 450, 460, 470, 480, 490, 500 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3200, 3300, 3400, 3500, 3600, 3700, 3800, 3900, 4000, 4100, 4200, 4300, 4400, 4500, 4600, 4700, 4800, 4900, 5000, 5100, 5200, 5300, 5400, 5500, 5600, 5700, 5800, 5900, 6000, 6100, 6200, 6300, 6400, 6500, 6600, 6700, 6800, 6900, 7000, 7100, 7200, 7300, 7400, 7500, 7600, 7700, 7800, 7900, 8000, 8100, 8200, 8300, 8400, 8500, 8600, 8700, 8800, 8900, 9000, 9100, 9200, 9300, 9400, 9500, 9600, 9700, 9800, 9900, 10000, 11000, 12000, 13000, 14000, 15000, 16000, 17000, 18000, 19000, 20000, 21000, 22000, 23000, 24000, 25000, 26000, 27000, 28000, 29000, 30000, 31000, 32000, 33000, 34000, 35000, 36000, 37000, 38000, 39000, 40000, 41000, 42000, 43000, 44000, 45000, 46000, 47000, 48000, 49000, 50000, 52500, 55000, 57500, 60000, 62500, 65000, 67500, 70000, 72500, 75000, 77500, 80000, 82500, 85000, 87500, 90000, 92500, 95000, 97500, 100000, 125000, 150000, 175000, 200000, 225000, 250000, 275000, 300000, 325000, 350000, 375000, 400000, 425000, 450000, 475000, 500000, 600000, 700000, 800000, 900000 или 1000000 последовательностей пептидов-ловушек. В некоторых вариантах осуществления по меньшей мере 20 тестовых пептидных последовательностей включают по меньшей мере 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200, 210, 220, 230, 240, 250, 260, 270, 280, 290, 300, 310, 320, 330, 340, 350, 360, 370, 380, 390, 400, 410, 420, 430, 440, 450, 460, 470, 480, 490, 500 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2100, 2200, 2300, 2400, 2500, 2600, 2700, 2800, 2900, 3000, 3100, 3200, 3300, 3400, 3500, 3600, 3700, 3800, 3900, 4000, 4100, 4200, 4300, 4400, 4500, 4600, 4700, 4800, 4900, 5000, 5100, 5200, 5300, 5400, 5500, 5600, 5700, 5800, 5900, 6000, 6100, 6200, 6300, 6400, 6500, 6600, 6700, 6800, 6900, 7000, 7100, 7200, 7300, 7400, 7500, 7600, 7700, 7800, 7900, 8000, 8100, 8200, 8300, 8400, 8500, 8600, 8700, 8800, 8900, 9000, 9100, 9200, 9300, 9400, 9500, 9600, 9700, 9800, 9900, 10000, 11000, 12000, 13000, 14000, 15000, 16000, 17000, 18000, 19000, 20000, 21000, 22000, 23000, 24000, 25000, 26000, 27000, 28000, 29000, 30000, 31000, 32000, 33000, 34000, 35000, 36000, 37000, 38000, 39000, 40000, 41000, 42000, 43000, 44000, 45000, 46000, 47000, 48000, 49000, 50000, 52500, 55000, 57500, 60000, 62500, 65000, 67500, 70000, 72500, 75000, 77500, 80000, 82500, 85000, 87500, 90000, 92500, 95000, 97500, 100000, 125000, 150000, 175000, 200000, 225000, 250000, 275000, 300000, 325000, 350000, 375000, 400000, 425000, 450000, 475000, 500000, 600000, 700000, 800000, 900000 или 1000000 тестовых пептидных последовательностей. В некоторых вариантах осуществления идентификация или назначение наивысшего процента из множества тестовых пептидных последовательностей в качестве презентируемых аллелем HLA класса II клетки включает идентификацию или назначения наивысших 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19%, 20%, 21%, 22%, 23%, 24%, 25%, 26%, 27%, 28%, 29%, 30%, 31%, 32%, 33%, 34%, 35%, 36%, 37%, 38%, 39%, или 40% в качестве презентируемых аллелем HLA класса II клетки. В некоторых вариантах осуществления клетка представляет собой моноаллельную клетку.
Система лейкоцитарного антигена человека (HLA)
[0435] Иммунную систему можно подразделить на две функциональных подсистемы: врожденная и адаптивная иммунная система. Врожденная иммунная система представляет собой первую линию защиты против инфекций, и большинство потенциальных патогенов быстро нейтрализуются этой системой до того, как они могут вызвать, например, заметную инфекцию. Адаптивная иммунная система реагирует с молекулярными структурами, называемыми антигенами, вторгающегося организма. В отличие от врожденной иммунной системы, адаптивная иммунная система является в высокой степени специфичной к патогену. Адаптивный иммунитет также может обеспечивать долговременную защиту; например, кто-либо, выздоровевший от кори, защищен от кори до конца его жизни. Существует два типа адаптивных иммунных реакций, которые включают гуморальную иммунную реакцию и клеточно-опосредуемую иммунную реакцию. При гуморальной иммунной реакции антитела, секретируемые B-клетками в жидкости организма, связываются с происходящими из патогенов антигенами, что приводит к устранению патогена посредством различных механизмов, например, опосредуемого комплементом лизиса. При клеточно-опосредуемой иммунной реакции активируются T-клетки, способные разрушать другие клетки. Например, если в клетке присутствуют белки, ассоциированные с заболеванием, они протеолитически фрагментируются на пептиды в клетке. Затем конкретные клеточные белки связываются с антигеном или пептидом, образовавшимся таким образом, и транспортируют их на поверхность клетки, где они презентируются молекулярным механизмам защиты в T-клетках организма. Цитотоксические T-клетки распознают эти антигены и уничтожают клетки, которые содержат антигены.
[0436] Термин "главный комплекс гистосовместимости (MHC)", "молекулы MHC" или "белки MHC" относится к белкам, способным связывать пептиды, образовавшиеся в результате протеолитического расщепления белковых антигенов и представляющие собой потенциальные T-клеточные эпитопы, транспортировать их на поверхность клетки и презентировать пептиды конкретным клеткам, например, цитотоксическим T-лимфоцитам или T-хелперным клеткам. MHC человека также называют комплексом HLA. Таким образом, термин "система лейкоцитарных антигенов человека (HLA)", "молекулы HLA" или "белки HLA" относится к генному комплексу, кодирующему белки MHC у человека. Термин MHC называют комплексом "H-2" в видах животных семейства мышиных. Специалистам в данной области будет понятно, что термины "главный комплекс гистосовместимости (MHC)", "молекулы MHC", "белки MHC" и "система лейкоцитарных антигенов человека (HLA)", "молекулы HLA", "белки HLA" используются в настоящем описании взаимозаменяемо.
[0437] Белки HLA подразделяют на два типа, обозначаемых как HLA класса I и HLA класса II. Структуры белков двух классов HLA являются в высокой степени сходными; однако они имеют в высокой степени различающиеся функции. Белки HLA класса I присутствуют на поверхности практически всех клеток организма, в том числе большинства опухолевых клеток. Белки HLA класса I нагружаются антигенами, которые обычно происходят из эндогенных белков или из патогенов, находящихся внутри клеток, а затем они презентируются наивным и цитотоксическим T-лимфоцитам (CTL). Белки HLA класса II присутствуют на антигенпредставляющих клетках (APC), включая, но не ограничиваясь ими, дендритные клетки, B-клетки и макрофаги. Они в основном презентируют пептиды, которые процессируются из внешних источников антигенов, например, извне клеток, хелперным T-клеткам. Большинство пептидов, связываемых белками HLA класса I, происходят из цитоплазматических белков, продуцируемых в здоровых клетках-хозяевах самого организма, и обычно не стимулируют иммунную реакцию.
[0438] Молекулы HLA класса I (фиг. 1) состоят из двух нековалентно связанных полипептидных цепей, HLA-кодируемой α-цепи (тяжелая цепь, от 44 до 47 кДа) и кодируемой не HLA субъединицы, называемой β2-микроглобулином (или β2m) (12 кДа). Цепь α имеет три внеклеточных домена: α1, α2 и α3 и трансмембранную область, среди которых области α1 и α2 способны связывать пептид приблизительно из 7-13 аминокислот (например, приблизительно от 8 до 11 аминокислоты, или 9 или 10 аминокислот). Молекула HLA класса 1 связывается с пептидом, который имеет подходящие связывающие мотивы, и презентирует его цитотоксическим T-лимфоцитам. Тяжелые цепи HLA класса 1 могут представлять собой белковый продукт аллеля HLA-A, также называемый мономером HLA-A, или белковый продукт аллеля HLA-B (аналогично, мономер HLA-B) или белковый продукт аллеля HLA-C (мономер HLA-C), каждый из которых образует комплексы с β-2-микроглобулином. α1 находится на белке не HLA β2m; β2m кодируется геном бета-2-микроглобулина, находящемся на хромосоме 15 человека. Домен α3 соединен с трансмембранной областью, заякоривающей молекулу HLA класса I на клеточной мембране. Презентируемый пептид удерживается дном связывающей пептид бороздки в центральной области гетеродимера α1/α2 (молекула, состоящая из двух неидентичных субъединиц). HLA класса I-A, HLA класса I-B или HLA класса I-C являются в высокой степени полиморфными. Каждый ген HLA класса 1-A (называемый геном HLA-A), ген HLA класса 1-B (называемый геном HLA-B) и ген HLA класса 1-C (называемый геном HLA-C) содержит 8 экзонов, экзон 1 кодирует лидерный пептид, экзоны 2 и 3 кодируют домены α1 и α2, экзон 5 кодирует трансмембранную область, и экзоны 6 и 7 кодируют цитоплазматическую хвостовую часть. Полиморфизмы экзона 2 и экзона 3 ответственны за специфичность связывания каждой молекулы класса 1. Ген HLA класса I-B (HLA-B) имеет множество возможных вариантов, паттернов экспрессии и презентируемых антигенов. Эта группа подразделяется на группу, кодируемую локусами HLA, например, HLA-E, HLA-F, HLA-G, а также группу, не кодируемую ими, например, лиганды стресса, такие как ULBP, Rae1 и H60. Антиген/лиганд для многих из этих молекул остается неизвестным, однако они могут взаимодействовать с любыми из CD8+ T-клеток, NKT-клеток и NK-клеток.
[0439] В некоторых вариантах осуществления в рамках настоящего изобретения используется неклассический аллель HLA класса I-E. Молекулы HLA-E распознаются натуральными киллерами (NK) и CD8+ T-клетками. HLA-E экспрессируется практически во всех тканях, включая легкое, печень, кожу и клетки плаценты. Экспрессия HLA-E также обнаруживается в солидных опухолях (например, остеосаркома и меланома). Молекула HLA-E связывается TCR, экспрессируемым на CD8+ T-клетках, что приводит к активации T-клеток. Также известно, что HLA-E связывает рецептор CD94/NKG2, экспрессируемый на NK-клетках и CD8+ T-клетках. CD94 может образовывать пару с несколькими различными изоформами NKG2, образуя рецепторы с потенциалом либо к ингибированию (NKG2A, NKG2B), либо к стимуляции (NKG2C) клеточной активации. HLA-E может связываться с пептидом, происходящим из аминокислотных остатков 3-11 лидерных последовательностей большинства молекул HLA-A, -B, -C и -G, однако он не может связываться с его собственным лидерным пептидом. Также было показано, что HLA-E презентирует пептиды, происходящие из эндогенных белков, сходных с аллелями HLA-A, -B и -C. В физиологических условиях встреча CD94/NKG2A с HLA-E, нагруженным пептидами из лидерных последовательностей HLA класса I, обычно индуцирует ингибиторные сигналы. Цитомегаловирус (CMV) использует этот механизм для ускользания от иммунного надзора NK-клетками посредством экспрессии гликопротеина UL40, имитирующего лидерную последовательность HLA-A. Однако также описано, что CD8+ T-клетки могут распознавать HLA-E, нагруженный пептидом UL40, происходящим из CMV штамма Toledo, и они участвуют в защите против CMV. Ряд исследований продемонстрировал несколько важных функций HLA-E при инфекционных заболеваниях и злокачественной опухоли.
[0440] Пептидные антигены связываются с молекулами HLA класса I посредством конкурентного аффинного связывания в эндоплазматической сети перед их презентацией на клеточной поверхности. В данном случае, аффинность индивидуального пептидного антигена прямо связана с его аминокислотной последовательностью и присутствием определенных связывающих мотивов в определенных положениях аминокислотной последовательности. Если последовательность такого пептида известна, можно манипулировать иммунной системой в отношении пораженных заболеванием клеток с использованием, например, пептидных вакцин.
[0441] Молекулы MHC являются в высокой степени полиморфными, т.е. существует множество вариантов MHC. Каждый вариант кодируется вариантом гена, кодирующего белок, и каждый такой вариант гена называется аллелем. Для человека MHC известен как лейкоцитарные антигены человека (HLA), которые включают три типа молекул HLA класса II: DP, DQ и DR. Пептиды HLA класса II (фиг. 1) имеют две цепи, α и β, каждая из которых имеет два домена- α1 и α2 и β1 и β2 - причем каждая цепь имеет трансмембранный домен, α2 и β2, соответственно, заякоривающий молекулу HLA класса II на клеточной мембране. Связывающая пептид бороздка образуется из гетеродимера α1 и β1. Наиболее широко исследованные молекулы HLA-DR имеют DRA и DRB, соответствующие доменам α и β, соответственно. DRB являются разнообразными, DRA практически идентичны. Таким образом, специфичность связывания аллеля DRB указывает на специфичность связывания соответствующего HLA-DR. Каждый белок MHC имеет свою собственную специфичность связывания, что означает, что набор пептидов, связывающихся с молекулой MHC, может отличаться от набора пептидов, связывающихся с другой молекулой MHC. Классические молекулы презентируют пептиды CD4+ лимфоцитам. Неклассические молекулы, являющиеся вспомогательными, с внутриклеточными функциями, не экспонируются на клеточных мембранах, а экспонируются на внутренних мембранах в лизосомах, обычно загружая антигенные пептиды на классические молекулы HLA класса II.
[0442] В системе HLA класса II фагоциты, такие как макрофаги и незрелые дендритные клетки, захватывают объекты посредством фагоцитоза в фагосомы - хотя B-клетки демонстрируют более общий эндоцитоз в эндосомы - которые сливаются с лизосомами, чьи кислые ферменты расщепляют захваченный белок на множество различных пептидов. Аутофагия является другим источником пептидов HLA класса II. Посредством физико-химической динамики молекулярного взаимодействия с образующимися у хозяина вариантами HLA класса II, кодируемыми геномом хозяина, конкретный пептид демонстрирует доминирование антигенной специфичности и загружается на молекулы HLA класса II. Они транспортируются и выводятся на клеточную поверхность. Большинство исследованных подклассов генов HLA класса II представляют собой: HLA-DPA1, HLA-DPB1, HLA-DQA1, HLA-DQB1, HLA-DRA и HLA-DRB1.
[0443] Презентация пептидов молекулами HLA класса II CD4+ хелперным T-клеткам необходима для иммунных ответов на чужеродные антигены (Roche and Furuta, 2015). После активации CD4+ T-клетки стимулируют дифференцировку B-клеток продуцирование антител, а также ответы CD8+ T-клеток (CTL). CD4+ T-клетки также секретируют цитокины и хемокины, которые активируют и индуцируют дифференцировку других иммунных клеток. Молекулы HLA класса II представляют собой гетеродимеры α- и β-цепей, которые взаимодействуют с образованием связывающей пептид бороздки, которая является более открытой, чем пептид-связывающие бороздки HLA класса I (Unanue et al., 2016). Полагают, что пептиды, связанные с молекулами HLA класса II, имеют связывающую центральную часть из 9 аминокислот с фланкирующими остатками либо с N-, либо с C-концевой стороны, которые выступают из бороздки (Jardetzky et al., 1996; Stern et al., 1994). Эти пептиды обычно имеют длину 12-16 аминокислот и часто содержат 3-4 якорных остатка в положениях P1, P4, P6/7 и P9 перечня связывающихся пептидов (Rossjohn et al., 2015).
[0444] Аллели HLA экспрессируются кодоминантным образом, что означает, что аллели (варианты), наследуемые от обоих родителей, экспрессируются в равной степени. Например, каждый индивидуум имеет 2 аллеля каждого из 3 генов класса I (HLA-A, HLA-B и HLA-C) и, таким образом, может экспрессировать шесть различных типов HLA класса II. В локусе HLA класса II каждый индивидуум наследует пару генов HLA-DP (DPA1 и DPB1, которые кодируют цепь α и β), HLA-DQ (DQA1 и DQB1, для цепей α и β), один ген HLA-DRα (DRA1), и один или более генов HLA-DRβ (DRB1 и DRB3, -4 или -5). HLA-DRB1, например, имеет более 400 известных аллелей. Это означает, что один гетерозиготный индивидуум может наследовать шесть или восемь функционирующих аллелей HLA класса II: по три или более от каждого родителя. Таким образом, гены HLA являются в высокой степени полиморфными; у различных индивидуумов внутри индивидуумов существует множество различных аллелей. Гены, кодирующие белки HLA, имеют множество различных вариантов, позволяя иммунной системе каждого индивидуума реагировать с широким диапазоном вторгшихся чужеродных агентов. Некоторые гены HLA имеют сотни идентифицированных версий (аллелей), каждому из которых присвоен конкретный номер. В некоторых вариантах осуществления аллели HLA класса I представляют собой HLA-A*02:01, HLA-B*14:02, HLA-A*23:01, HLA-E*01:01 (неклассические). В некоторых вариантах осуществления аллели HLA класса II представляют собой HLA-DRB*01:01, HLA-DRB*01:02, HLA-DRB*11:01, HLA-DRB*15:01 и HLA-DRB*07:01.
[0445] Специфические для индивидуума аллели HLA или генотип HLA индивидуума можно определять любым способом, известным в данной области. В иллюстративных вариантах осуществления генотипы HLA определяют любым способом, описанным в международной патентной заявке номер PCT/US2014/068746, опубликованной 11 июня 2015 года в качестве W02015085147, которая включена в настоящее описание в качестве ссылки в полном объеме. В кратком изложении способы включают определение типов полиморфных генов, которое может включать проведение выравнивания чтений, извлеченных из набора данных секвенирования, с эталонным набором генов, содержащим варианты аллелей полиморфного гена, определение первой апостериорной вероятности или показателя, установленного из апостериорной вероятности, для каждого аллельного варианта в выравнивании, идентификацию аллельного варианта с максимальной первой апостериорной вероятностью или показателем, установленным из апостериорной вероятности, в качестве первого аллельного варианта, идентификацию одного или более перекрывающихся чтений, выровненных с первым аллельным вариантом и одним или более другими аллельными вариантами, определение второй апостериорной вероятности или показателя, установленного из апостериорной вероятности, для одного или более других аллельных вариантов с использованием коэффициента взвешивания, идентификацию второго аллельного варианта посредством выбора аллельного варианта с максимальной второй апостериорной вероятностью или показателем, установленным из апостериорной вероятности, где первый и второй аллельные варианты определяют тип гена для полиморфного гена, и предоставление результата для первого и второго аллельного варианта.
[0446] В некоторых вариантах осуществления способы прогнозирования связывания и презентации пептид MHC класса II: антигенный пептид, описанные в настоящем описании, способны прогнозировать связывающие соединения из большого репертуара пептидов MHC класса II, кодируемых индивидуальными аллелями HLA. В некоторых вариантах осуществления технологию MAPTAC обучают с использованием большой базы данных валидированных посредством масс-спектрометрии соответствующих HLA пептидов. В некоторых вариантах осуществления большая база данных валидированных посредством масс-спектрометрии соответствующих HLA пептидов включает более 1,2×10^6 таких соответствующих HLA пептидов. В некоторых вариантах осуществления большая база данных валидированных посредством масс-спектрометрии соответствующих HLA пептидов охватывает более 150 аллелей HLA, включающих подтипы аллелей MHC класса I и класса II. В некоторых вариантах осуществления база данных охватывает по меньшей мере 95% популяции США в отношении HLA-I и HLA-II (подтип DR).
[0447] Как описано в настоящем описании существуют убедительные доказательства как у животных, так и у человека, что мутантные эпитопы являются эффективными в отношении индукции иммунного ответа и что случаи спонтанной регрессии опухоли или долговременного выживания коррелируют с CD8+ T-клеточными ответами на мутантные эпитопы, и что "иммунное редактирование" может отслеживаться до изменений экспрессии доминантных мутантных антигенов у мышей и человека.
[0448] Технология секвенирования продемонстрировала, что каждая опухоль содержит множество специфических для пациента мутаций, которые изменяют кодирующее белок содержимое гена. Такие мутации создают измененные белки в диапазоне от единичных аминокислотных изменений (вызываемых миссенс-мутациями) до вставок длинных участков новых аминокислотных последовательностей вследствие сдвига рамки считывания, сквозного прохождения кодонов терминации или трансляции интронных областей (новые мутации открытых рамок считывания; neoORF). Эти мутантные белки являются ценными мишенями для иммунного ответа хозяина на опухоль, поскольку, в отличие от нативных белков, они не подвергаются ослабляющим иммунитет эффектам толерантности к своему. Таким образом, мутантные белки более вероятно будут иммуногенными и также являются более специфическими в отношении опухолевых клеток по сравнению с нормальными клетками пациента. В сущности, короткие пептиды (длиной 8-24 аминокислоты), содержащие ассоциированные со злокачественной опухолью мутацию, являются кандидатами для иммунотерапии злокачественной опухоли.
[0449] В некоторых вариантах осуществления алгоритм, лежащий в основе способа прогнозирования, можно далее использовать для идентификации мутаций пептида. В некоторых вариантах осуществления способ прогнозирования можно использовать для определения статуса драйверной мутации, и/или статуса экспрессии РНК, и/или прогнозирования расщепления в пептиде.
[0450] Термин "T-клетка" включает CD4+ T-клетки и CD8+ T-клетки. Также термин "T-клетка" включает как T-хелперные клетки 1 типа и T-хелперные клетки 2 типа. T-клетки, как используют в рамках изобретения, главным образом, классифицируют в зависимости от функции и антигенов клеточной поверхности (антигены кластерной дифференцировки, или CD), которые также способствуют связыванию T-клеточного рецептора с антигеном, на два основных класса: хелперные T-клетки (TH) и цитотоксические T-лимфоциты (CTL).
[0451] Зрелые хелперные T-клетки (TH) экспрессируют поверхностный белок CD4, и их обозначают как CD4+ T-клетки. После развития T-клеток созревшие наивные T-клетки покидают тимус и начинают распространяться по организму, включая лимфатические узлы. Наивные T-клетки представляют собой T-клетки, которые никогда не были экспонированы антигену, на который они запрограммированы отвечать. Подобно всем T-клеткам, они экспрессируют комплекс T-клеточный рецептор-CD3. T-клеточный рецептор (TCR) состоит как из константных, так и из вариабельных областей. Вариабельная область определяет, на какой антиген T-клетка может отвечать. CD4+ T-клетки имеют TCR с аффинностью в отношении белков MHC класса II, и он вовлечен в определение аффинности к MHC в ходе созревания в тимусе. Белки MHC класса II, как правило, находятся на поверхности специализированных антигенпредставляющих клеток (APC). Специализированные антигенпредставляющие клетки (APC) в основном представляют собой дендритные клетки, макрофаги и B-клетки, хотя дендритные клетки являются единственной группой клеток, которые конститутивно экспрессируют MHC класса II (во все моменты времени). Некоторые APC также связывают нативные (или непроцессированные) антигены с их поверхностью, такие как фолликулярные дендритные клетки, однако непроцессированные антигены не взаимодействуют с T-клетками и не вовлечены в их активацию. Пептидные антигены, которые связываются с белками HLA класса I, как правило, короче, чем пептидные антигены, которые связываются с белками HLA класса II.
[0452] Цитотоксические T-лимфоциты (CTL), также известные как цитотоксические T, цитолитические T-клетки, CD8+ T-клетки или киллерные T-клетки, относятся к лимфоцитам, которые индуцируют апоптоз в клетках-мишенях. CTL формируют антигенспецифические конъюгаты с клетками-мишенями посредством взаимодействия TCR с процессированным антигеном (Ag) на поверхностях клеток-мишеней, что приводит к апоптозу клетки-мишени. Апоптотические тела удаляются макрофагами. Термин "ответ CTL" используют для указания на первичный иммунный ответ, опосредуемый клетками CTL. Цитотоксические T-лимфоциты имеют как T-клеточные рецепторы (TCR), так и молекулы CD8, на их поверхности. T-клеточные рецепторы способны распознавать и связывать пептиды в комплексе с молекулами HLA класса I. Каждый цитотоксический T-лимфоцит экспрессирует уникальный T-клеточный рецептор, который способен связывать определенные комплексы MHC/пептид. Большинство цитотоксических T-клеток экспрессируют T-клеточные рецепторы (TCR), которые могут распознавать специфический антиген. Для связывания TCR с молекулой HLA класса I они должны сопровождаться гликопротеином, называемым CD8, который связывается с константной частью молекулы HLA класса I. Таким образом, эти T-клетки называются CD8+ T-клетками. Аффинность между CD8 и молекулой MHC удерживает T-клетку и клетку-мишень тесно связанными в ходе антиген-специфической активации. CD8+ T-клетки распознаются в качестве T-клеток после активации и, как правило, их определяют как имеющие предопределенную цитотоксическую роль в иммунной системе. Однако CD8+ T-клетки также способны продуцировать некоторые цитокины.
[0453] "T-клеточные рецепторы (TCR)" представляют собой рецепторы клеточной поверхности, которые участвуют в активации T-клеток в ответ на презентацию антигена. TCR, главным образом, состоит из двух цепей, альфа и бета, которые собираются с образованием гетеродимера, и он связан с CD3-трансдуцирующими субъединицами с образованием T-клеточного рецепторного комплекса, находящегося на клеточной поверхности. Каждая цепь альфа и бета в TCR состоит из иммуноглобулин-подобной N-концевой вариабельной (V) и константной (C) области, гидрофобного трансмембранного домена и короткой цитоплазматической области. Что касается молекул иммуноглобулинов, вариабельные области альфа- и бета-цепей образуются посредством V(D)J-рекомбинации, создавая большое разнообразие антигенной специфичности в популяции T-клеток. Однако в противоположность иммуноглобулинам, которые распознают интактный антиген, T-клетки активируются процессированными пептидными фрагментами, связанными с молекулой MHC, обеспечивающей дополнительный аспект распознавания антигена T-клетками, известный как MHC-рестрикция. Распознавание несоответствий MHC между донором и реципиентом посредством T-клеточного рецептора приводит к пролиферации T-клеток и потенциальному развитию GVHD. Было показано, что нормальная поверхностная экспрессия TCR зависит от скоординированного синтеза и сборки всех семи компонентов комплекса (Ashwell and Klusner 1990). Инактивация TCRα или TCRβ может приводить к устранению TCR с поверхности T-клеток, препятствуя распознаванию аллоантигена и, таким образом, GVHD. Однако, разрушение TCR, как правило, приводит к устранению компонента передачи сигнала CD3 и изменяет способы дальнейшей T-клеточной экспансии.
[0454] Термин "HLA-пептидом" относится к совокупности пептидов, которые специфически взаимодействуют с конкретным классом HLA и могут охватывать тысячи различных последовательностей. Пептидомы HLA включают многообразие пептидов, происходящих как из нормальных, так и из аномальных белков, экспрессируемых в клетках. Таким образом, пептидомы HLA можно исследовать для идентификации специфических для злокачественной опухоли пептидов, для разработки средств иммунотерапии злокачественной опухоли и в качестве источника информации о схемах синтеза и деградации белков в злокачественных клетках. В некоторых вариантах осуществления пептидом HLA представляет собой совокупность растворимых пептидов HLA (sHLA). В некоторых вариантах осуществления пептидом HLA представляет собой совокупность ассоциированного с мембраной HLA (mHLA).
[0455] "Антигенпрезентирующая клетка" или "APC" включает профессиональные антигенпредставляющие клетки (например, B-лимфоциты, макрофаги, моноциты, дендритные клетки, клетки Лангерганса), а также другие антигенпредставляющие клетки (например, кератиноциты, эндотелиальные клетки, астроциты, фибробласты, олигодендроциты, тимические эпителиальные клетки, эпителиальные клетки щитовидной железы, глиальные клетки (головной мозг), бета-клетки поджелудочной железы и эндотелиальные клетки сосудов). "Антигенпредставляющая клетка" или "APC" представляет собой клетку, которая экспрессирует молекулы главного комплекса гистосовместимости (MHC) и может экспонировать чужеродный антиген в комплексе с MHC на ее поверхности.
Моноаллельные по HLA клеточные линии
[0456] Моноаллельную клеточную линию, экспрессирующую либо единичный аллель HLA класса I, либо одну пару аллелей HLA класса II, либо единичный аллель HLA класса I, либо одну пару аллелей HLA класса II, можно получать посредством трансдукции или трансфекции подходящей клеточной популяции посредством полинуклеиновой кислоты, например, вектора, кодирующего единичный аллель HLA (фиг. 2). Подходящие клеточные популяции включают, например, клеточные линии с дефектом HLA класса I, в которых единичный аллель HLA класса I экзогенно экспрессируется, клеточные линии с дефектом HLA класса II, в которых экспрессируется одна экзогенная пара аллелей HLA класса II, или клеточные линии с дефектом класса I и класса II, в которых единичный аллель HLA класса I и/или одна пара аллелей класса II экзогенно экспрессируется. В качестве иллюстративного варианта осуществления B-клеточная линия с дефектом HLA класса I представляет собой B721.221. Однако специалисту в данной области понятно, что можно получать другие клеточные популяции, которые имеют дефект HLA класса I и/или HLA класса II. Иллюстративный способ делеции/инактивации эндогенных генов HLA класса I или HLA класса II включает опосредуемое CRISPR-Cas9 редактирование генома, например, в клетках THP-1. В некоторых вариантах осуществления популяции клеток представляют собой профессиональные антигенпредставляющие клетки, такие как макрофаги, B-клетки и дендритные клетки. Клетки могут представлять собой B-клетки или дендритные клетки. В некоторых вариантах осуществления клетки представляют собой опухолевые клетки или клетки из опухолевой клеточной линии. В некоторых вариантах осуществления клетки выделяют от пациента. В некоторых вариантах осуществления клетки содержат инфекционный агент или его часть. В некоторых вариантах осуществления популяция клеток содержит по меньшей мере 107 клеток. В некоторых вариантах осуществления популяцию клеток далее модифицируют, например, посредством повышения или снижения экспрессии и/или активности по меньшей мере одного гена. В некоторых вариантах осуществления ген кодирует член иммунопротеасомы. Известно, что иммунопротеасома вовлечена в процессинг пептидов, связывающих HLA класса I, и включает субъединицы LMP2 (β1i), MECL-1 (β2i) и LMP7 (β5i). Иммунопротеасома также может индуцироваться интерфероном-гамма. Таким образом, в некоторых вариантах осуществления популяцию клеток можно приводить в контакт с одним или более цитокинами, факторами роста или другими белками. Клетки можно стимулировать воспалительными цитокинами, такими как интерферон-гамма, IL-10, IL-6 и/или TNF-α. Популяцию клеток также можно подвергать различным условиям внешней среды, таким как стресс (тепловой стресс, лишение кислорода, глюкозное голодание, повреждающие ДНК средства и т.д.). В некоторых вариантах осуществления клетки приводят в контакт с одним или более химиотерапевтическими средствами, лучевой терапией, направленной терапией или иммунотерапией. Таким образом, способы, описанные в настоящем описании, могут использоваться для исследования эффекта различных генов или условий на процессинг и презентацию пептидов посредством HLA. В некоторых вариантах осуществления используемые условия выбирают таким образом, чтобы они соответствовали состоянию пациента, для которого намереваются идентифицировать популяцию HLA-пептидов.
[0457] Единичный аллель HLA по настоящему изобретению может кодироваться и экспрессироваться с использованием системы на вирусной основе (например, аденовирусная система, аденоассоциированный вирусный (AAV) вектор, поксвирус или лентивирус). Плазмиды, которые могут использоваться для доставки посредством аденоассоциированного вируса, аденовируса и лентивируса, описаны ранее (см., например, патенты США №6955808 и 6943019, и патентную заявку США №20080254008, включенную в настоящее описание в качестве ссылки). Среди векторов, которые могут применяться на практике в рамках настоящего изобретения, является возможным встраивание в геном клетки-хозяина с использованием ретровирусных способов переноса генов, что часто приводит к долговременной экспрессии встроенного трансгена. В иллюстративном варианте осуществления ретровирус представляет собой лентивирус. Кроме того, высокую эффективность трансдукции наблюдают во многих различных типах клеток и тканях-мишенях. Тропизм ретровируса может быть изменен путем включения чужеродных белков оболочки, расширяющего потенциальную популяцию клеток-мишеней. Ретровирус также можно конструировать, чтобы позволить зависимую от условий экспрессию встроенного трансгена, так чтобы только определенные типы клеток инфицировались лентивирусом. Специфические для типов клеток промоторы можно использовать для нацеливания на экспрессию в конкретных типах клеток. Лентивирусные векторы представляют собой ретровирусные векторы (и, таким образом, как лентивирусные, так и ретровирусные векторы могут использоваться для применения настоящего изобретения на практике). Более того, лентивирусные векторы способны трансдуцировать или инфицировать неделящиеся клетки и, как правило, продуцировать высокие титры вируса.
[0458] Выбор ретровирусной системы переноса генов может зависеть от ткани-мишени. Ретровирусные векторы включают цис-действующие длинные концевые повторы со способностью упаковывать вплоть до 6-10 т.п.н. чужеродной последовательности. Минимальные цис-действующие LTR являются достаточными для репликации и упаковки векторов, которые затем используют для встраивания желаемой нуклеиновой кислоты в клетку-мишень для обеспечения постоянной экспрессии. Широко используемые ретровирусные векторы, которые могут использоваться при применении настоящего изобретения на практике, включают ретровирусные векторы на основе вируса лейкоза мышей (MuLV), вируса лейкоза гиббонов (GaLV), вируса иммунодефицита обезьян (SIV), вируса иммунодефицита человека (ВИЧ) и их комбинаций (см., например, Buchscher et al., (1992) J. Virol. 66:2731-2739; Johann et al., (1992) J. Virol. 66:1635-1640; Sommnerfelt et al., (1990) Virol. 176:58-59; Wilson et al., (1998) J. Virol. 63:2374-2378; Miller et al., (1991) J. Virol. 65:2220-2224; PCT/US94/05700). Также для применения настоящего изобретения на практике является пригодным минимальный лентивирусный вектор не приматов, такой как лентивирусный вектор на основе вируса инфекционной анемии лошадей (EIAV) (см., например, Balagaan, (2006) J Gene Med; 8: 275-285, опубликована через интернет 21 ноября 2005 года в Wiley InterScience DOI: 10.1002/jgm.845). Векторы могут иметь промотор цитомегаловируса (CMV), контролирующий экспрессию гена-мишени. Таким образом, настоящее изобретение предусматривает, среди вектора(ов), пригодного для применения настоящего изобретения на практике: вирусные векторы, включая ретровирусные векторы и лентивирусные векторы.
[0459] Любой аллель HLA может экспрессироваться в клеточной популяции. В иллюстративном варианте осуществления аллель HLA представляет собой аллель HLA класса I. В некоторых вариантах осуществления аллель HLA класса I представляет собой аллель HLA-A или аллель HLA-B. В некоторых вариантах осуществления аллель HLA представляет собой аллель HLA класса II. Последовательности аллелей HLA класса I и класса II могут быть найдены в базе данных IPD-IMGT/HLA Database. Иллюстративные аллели HLA включают, но не ограничиваются ими, HLA-A*02:01, HLA-B*14:02, HLA-A*23:01, HLA-E*01:01, HLA-DRB*01:01, HLA-DRB*01:02, HLA-DRB*11:01, HLA-DRB*15:01 и HLA-DRB*07:01.
[0460] В некоторых вариантах осуществления аллель HLA выбирают так, чтобы он соответствовал представляющему интерес генотипу. В некоторых вариантах осуществления аллель HLA представляет собой мутантный аллель HLA, который может представлять собой не встречающийся в природе аллель или встречающийся в природе аллель у страдающего пациента. Способы, описанные в настоящем описании, имеют дополнительное преимущество идентификации связывающих HLA пептидов для аллелей HLA, ассоциированных с различными нарушениями, а также аллелей, которые присутствуют с низкой частотой. Таким образом, в некоторых вариантах осуществления способ, описанный в настоящем описании, может идентифицировать аллель HLA, даже если он присутствует с частотой менее 1% в популяции, такой как европиоидная популяция.
[0461] В некоторых вариантах осуществления последовательность нуклеиновой кислоты, кодирующая аллель HLA, дополнительно включает аффинную акцепторную метку, которая может использоваться для иммунной очистки белка HLA. Подходящие метки хорошо известны в данной области. В некоторых вариантах осуществления аффинная акцепторная метка представляет собой полигистидиновую метку, поли-гистидин-глициновую метку, поли-аргининовую метку, поли-аспартатную метку, поли-цистеиновую метку, поли-фенилаланин, c-myc-метку, метку на основе гликопротеина D (gD) вируса простого герпеса, FLAG-метку, эпитопную метку KT3, эпитопную метку тубулина, пептидную метку на основе белка 10 гена T7, стрептавидиновую метку, метку на основе стрептавидин-связывающего пептида (SPB), Strep-метку, Strep-метку II, метку на основе альбумин-связывающего белка (ABP), метку на основе щелочной фосфатазы (AP), метку на основе вируса синего языка (B-метка), метку на основе кальмодулин-связывающего пептида (CBP), метку на основе хлорамфениколацетилтрансферазы (CAT), метку на основе холин-связывающего домена (CBD), метку на основе хитин-связывающего домена (CBD), метку на основе связывающего целлюлозу домена (CBP), метку на основе дигидрофолатредуктазы (DHFR), метку на основе галактоза-связывающего белка (GBP), мальтоза-связывающий белок (MBP), глутатион-S-трансферазу (GST), метку Glu-Glu (EE), метку на основе гемагглютинина (HA) вируса гриппа, метку на основе пероксидазы хрена (HRP), NE-метку, HSV-метку, метку на основе кетостероидизомеразы (KSI), KT3-метку, LacZ-метку, люциферазную метку, NusA-метку, метку на основе PDZ-домена, AviTag, метку на основе кальмодулина, E-метку, S-метку, SBP-метку, Softag 1, Softag 3, TC-метку, VSV-метку, Xpress-метку, Isopeptag, SpyTag, SnoopTag, метку Profinity eXact, метку на основе белка C, S1-метку, S-метку, метку на основе белка-переносчика биотин-карбокси (BCCP), метку на основе зеленого флуоресцентного белка (GFP), метку на основе малого убиквитин-подобного модификатора (SUMO), метку для тандемной аффинной очистки (TAP), HaloTag, Nus-метку, тиоредоксиновую метку, Fc-метку, метку CYD, метку HPC, метку TrpE, убиквитиновую метку, эпитопную метку VSV-G, происходящую из гликопротеина вируса везикулярного стоматита или V5-метку, происходящую из малого эпитопа (Pk), присутствующего на белках P и V парамиксовируса, вируса обезьян 5 (SV5). В некоторых вариантах осуществления аффинная акцепторная метка представляет собой "эпитопную метку", которая представляет собой тип пептидной метки, который добавляет распознаваемый эпитоп (участок связывания антитела) к белку HLA, для обеспечения связывания соответствующего антитела, тем самым позволяя идентификацию или аффинную очистку меченого белка. Неограничивающим примером эпитопной метки является белок A или белок G, который связывается с IgG. В некоторых вариантах осуществления аффинные акцепторные метки включают последовательность биотин-акцепторного пептида (BAP) или гемагглютинина вируса гриппа (HA). Многочисленные другие метки известны и могут быть предусмотрены специалистам в данной области, и они охватываются настоящим изобретением. Может использоваться любая пептидная метка при условии, что она способна быть экспрессированной в качестве элемента меченного аффинной акцепторной меткой комплекса HLA-пептид.
[0462] Способы, описанные в настоящем описании, включают выделение комплексов HLA-пептид из клеток, трансфицированных или трансдуированных посредством аффинной адсорбции конструкций HLA (фиг. 3). В некоторых вариантах осуществления комплексы могут быть выделены с использованием стандартных способов иммунопреципитации, известных в данной области, с использованием коммерчески доступных антител. Сначала клетки можно лизировать. Комплексы HLA класса I-пептид можно выделять с использованием антител, специфичных к HLA класса I, таких как антитело W6/32, в то время как комплексы HLA класса II-пептид можно выделять с использованием антител, специфичных к HLA класса II, таких как моноклональное антитело M5/114.15.2. В некоторых вариантах осуществления единичные (или пара) аллели HLA экспрессируются в качестве слитого белка с пептидной меткой, и комплексы HLA-пептид выделяют с использованием связывающих молекул, которые распознают пептидные метки.
[0463] Кроме того, способы включают выделение пептидов из указанных комплексов HLA-пептид и секвенирование пептидов. Пептиды выделяют из комплекса любым способом, известным специалисту в данной области, таким как кислотное элюирование. В то время как можно использовать любой способ секвенирования, в некоторых вариантах осуществления используются способы с использованием масс-спектрометрии, такие как жидкостная хроматография-масс-спектрометрия (LC-MS или LC-MS/MS, или альтернативно ВЭЖХ-MS или ВЭЖХ-MS/MS). Эти способы секвенирования хорошо известны специалисту в данной области и рассмотрены в Medzihradszky KF and Chalkley RJ. Mass Spectrom Rev. 2015 Jan-Feb;34(1):43-63.
[0464] В некоторых вариантах осуществления популяция клеток экспрессирует один или более эндогенных аллелей HLA. В некоторых вариантах осуществления популяция клеток представляет собой модифицированную популяцию клеток, лишенных одного или более эндогенных аллелей HLA класса I. В некоторых вариантах осуществления популяция клеток представляет собой модифицированную популяцию клеток, лишенную эндогенных аллелей HLA класса I. В некоторых вариантах осуществления популяция клеток представляет собой модифицированную популяцию клеток, лишенную одного или более эндогенных аллелей HLA класса II. В некоторых вариантах осуществления популяция клеток представляет собой модифицированную популяцию клеток, лишенную эндогенных аллелей HLA класса II, или модифицированную популяцию клеток, лишенную эндогенных аллелей HLA класса I и эндогенных аллелей HLA класса II. В некоторых вариантах осуществления популяция клеток содержит клетки, которые обогащены или отсортированы, например, посредством активированной флуоресценцией сортировки клеток (FACS). В некоторых вариантах осуществления активированную флуоресценцией сортировку клеток (FACS) используют для сортировки популяции клеток. В некоторых вариантах осуществления популяция клеток предварительно отсортирована посредством FACS в отношении экспрессии на клеточной поверхности HLA либо класса I, либо класса II, или HLA как класса I, так и класса II. Например, FACS может использоваться для сортировки популяции клеток в отношении экспрессии на клеточной поверхности аллеля HLA класса I, аллеля HLA класса II, или их комбинации.
Способы получения персонализированной вакцины против злокачественной опухоли
[0465] После идентификации мутации, специфической для злокачественной опухоли, так что мутация существует в ДНК злокачественных клеток, но не нормальных клетках того же человека и мутация приводит к замене одной или более аминокислот в белке, кодируемом ДНК, мутация может стать мишенью для иммунного ответа хозяина. Естественный иммунный ответ может быть направлен против мутантного белка, что приводит к разрушению злокачественных клеток, экспрессирующих белок. Вследствие естественного ответа толерантности и иммунодефицитной среды в злокачественной ткани, иммунотерапия является клиническим путем, который направлен на усиление такого иммунного ответа для преодоления толерантности и иммуносупрессивных эффектов организма. Таким образом, белок или пептид, содержащий мутацию, как описано выше, является подходящим кандидатом для иммунотерапии.
[0466] Мутантный белок поглощается профессиональными фагоцитами, выступающими в качестве антигенпредставляющих клеток (APC), разрезается и экспонируется в качестве антигенов на клеточной поверхности для активации T-клеток в антигенпрезентирующем комплексе, содержащем белок главного комплекса гистосовместимости (MHC). Белки MHC человека называют лейкоцитарными антигенами человека, HLA. Белок MHC может представлять собой белок MHC класса I или класса II, и, хотя несколько функциональных особенностей свойственно презентации пептидов белками MHC либо класса I, либо класса II (белки HLA класса I и HLA класса II), одна важная особенность состоит в том факте, что комплексы HLA класса I-пептид презентируют антигены цитотоксическим CD8+ T-клеткам, в то время как комплексы HLA класса II-пептид также способны активировать CD4+ T-клетки, что приводит к более длительному иммунному ответу. CD8+ T-клетки незаменимы в задаче устранения клетки клеткой для пораженных заболеванием клеток, таких как инфицированные клетки или опухолевые клетки. CD4+ T-клетки имеют более длительные эффекты при активации, причем наиболее важным из них является формирование иммунологической памяти. Подгруппы CD4 по-разному привлекаются в зависимости от типа иммунологической угрозы, и также могут совместно привлекаться множественные подгруппы с перекрывающимися или отличающимися функциями. Это помогает уравновешивать иммунологический ответ в отношении патогенной угрозы. В этих отношениях опосредуемая пептидом HLA класса II презентация антигена обеспечивает длительный и адаптированный иммунный ответ. С другой стороны, связывание HLA класса II с пептидами может быть неизбирательным и, таким образом, неспецифическое связывание и презентация иммунной системе пептидов приводит к аберрантному иммунному ответу, такому как аутоиммунитет.
[0467] В одном аспекте настоящее изобретение относится к способу прогнозирования пептидов, которые точно могут образовывать пару или связываться с определенным гетеродимером альфа- и бета-цепи HLA класса II, так что высокоточное связывание пептида с белком HLA класса II (содержащим гетеродимер альфа- и бета-цепи) обеспечивает презентацию специфического пептида T-лимфоцитам, тем самым индуцируя специфический иммунный ответ и избегая какой-либо перекрестной реактивности или иммунной неизбирательности.
[0468] В одном аспекте настоящее изобретение относится к способу прогнозирования пептидов, которые точно могут связываться с определенным белком HLA класса II, так что пептид может активировать более длительный и устойчивый иммунный ответ, когда пептид вводят терапевтически индивидууму, у которого экспрессируется определенный собственный белок HLA класса II, посредством усиления способности активации CD4+ T-клеток белком HLA класса II и стимуляции иммунологической памяти. В некоторых вариантах осуществления данный пептид, для которого спрогнозировано связывание с белком HLA класса II с высокой специфичностью, представляет собой пептид, содержащий мутацию, где мутация распространена в злокачественной опухоли или опухолевой клетке индивидуума; в то время как тот же белок HLA класса II, спрогнозированный в качестве связывающего мутантный пептид, либо (a) не связывается, или (b) связывается с заметно более низкой аффинностью с соответствующим немутантным пептидом дикого типа по сравнению с аффинностью для связывания с мутантным пептидом индивидуума. Предпочтительное связывание HLA с мутантным пептидом является преимущественным при разработке иммунотерапевтического средства, поскольку клетки, экспрессирующие пептид дикого типа, будут спасены от иммунной атаки T-клеток, реактивных к презентируемому посредством HLA пептиду. В некоторых вариантах осуществления спрогнозированные пептиды, которые специфически связываются с белками HLA класса II, представляют собой пептиды, которые имеют посттрансляционные модификации. Иллюстративные посттрансляционные модификации включают, но не ограничиваются ими: фосфорилирование, убиквитинилирование, дефосфорилирование, гликозилирование, метилирование или ацетилирование. В некоторых вариантах осуществления спрогнозированные пептиды подвергают посттрансляционным модификациям перед применением в иммунотерапии.
[0469] В некоторых вариантах осуществления способы и стратегии иммунотерапии, описанные в настоящем описании, также могут быть применимы для подавления нежелательной иммунной активации, например, в аутоиммунной реакции. В частности, пептиды, идентифицированные в качестве потенциальных связывающих молекул для определенных подтипов HLA, могут быть приспособлены для связывания с определенной молекулой HLA и индукции толерантности, а не обеспечения иммуногенного ответа.
[0470] В одном аспекте в настоящем описании описаны иммунотерапии, адаптированные или персонализированные для конкретного индивидуума. У каждого индивидуума или пациента экспрессируется определенный набор белков HLA класса I и HLA класса II. HLA-типирование представляет собой хорошо известный способ, который позволяет определение конкретного репертуара белков HLA, экспрессируемых у индивидуума. После того, как гетеродимеры HLA, экспрессируемые у конкретного индивидуума, известны, наличие усовершенствованного, продвинутого и надежного способа, как описано в настоящем описании, для прогнозирования пептидов, которые могут связываться с конкретным гетеродимером альфа- и бета-цепи HLA класса II с высокой точностью, может гарантировать индукцию специфического иммунного ответа, адаптированного конкретно для индивидуума.
[0471] Гены, кодирующие гетеродимеры HLA, являются в высокой степени полиморфными с более 4000 вариантов аллелей HLA класса II, идентифицированных в человеческой популяции. Из материнских и отцовских гаплотипов HLA индивидуум может наследовать различные аллели для каждого из локусов HLA класса II, и каждый гетеродимер HLA класса II состоит из α- и β-цепи. Вследствие большого количества комбинаций пар α- и β-цепей, особенно для аллелей HLA-DP и HLA-DQ, совокупность возможных гетеродимеров HLA является в высокой степени комплексной. Гетеродимеры HLA класса II транслируются в эндоплазматической сети (ER) и собираются в стабильный комплекс с инвариантной цепью (Ii), происходящей из белка CD74. Ii стабилизирует комплекс класса II, обеспечивая надлежащее сворачивание белков, и позволяет экспорт гетеродимеров HLA класса II в эндосомальные/лизосомальные компартменты. Внутри этих компартментов загрузки HLA класса II, Ii протеолитически расщепляется катепсинами до пептида-местозаполнителя, называемого CLIP. Затем CLIP заменяется на более высокоаффинные пептиды в среде с низким pH посредством шаперона HLA-DM, неклассического гетеродимера HLA класса II. Затем высокоаффинные нагруженные пептидом комплексы HLA класса II перемещаются в транс-Гольджи и, наконец, на клеточную поверхность для экспонирования CD4+ T-клеткам.
[0472] Согласно оценке, каждый гетеродимер HLA связывает тысячи пептидов с аллелеспецифическим предпочтением связывания. В действительности, согласно оценке, каждый аллель HLA связывает и презентирует ~1000-10000 уникальных пептидов T-клеткам. Учитывая такое разнообразие связывания HLA, точное прогнозирование вероятности связывания пептида с определенным аллелем HLA является в высокой степени затрудненным. Меньше известно об аллелеспецифических характеристиках связывания пептидов молекул HLA класса II вследствие гетерогенности образования пар α- и β-цепей, комплексности данных, ограничивающих возможность надежно назначать центральные связывающие эпитопы, и недостаточной степени иммунопреципитации, и об аллелеспецифических антителах, необходимых для биохимического анализа высокого разрешения. Более того, анализ пептидных эпитопов, происходящих из данного аллеля HLA, приводит к неопределенности, когда на клеточной поверхности презентируется несколько аллелей HLA.
[0473] Прогнозирование неоантигенов-кандидатов в основном проводится для эпитопов HLA класса I (учитывая доступность экспериментальных данных для алгоритмов прогнозирования класса I по сравнению с классом II), тем не менее ответы CD4+ T-клеток часто наблюдаются как в доклинических, так и в клинических испытаниях персонализированной вакцинации неоантигенами. Эти наблюдения демонстрируют, что процессинг и презентация эпитопа посредством HLA класса II также могут играть ключевую роль в лечении злокачественной опухоли. Хотя существуют алгоритмы прогнозирования для HLA класса II, они являются неточными, поскольку незамкнутая связывающая пептиды бороздка на гетеродимерах HLA класса II позволяет связывание более длинных пептидов (как правило, 15-40 аминокислот), что повышает гетерогенность и комплексность презентации эпитопов. Таким образом, необходима дальнейшая работа для лучшего понимания характеристик пептид-связывающих центров HLA класса II и клеточных процессов, вовлеченных в процессинг и презентацию эпитопов класса II. Область протеомики в настоящее время ограничена комплексностью образования гетеродимеров HLA класса II и доступностью антител для иммунопреципитации для выделения комплекса HLA класса II-пептид. Для решения этих задач был разработан процесс моноаллельного определения профиля HLA, который основан на LC-MS/MS для охарактеризации аллелеспецифических лигандомов для HLA класса II для способов прогнозирования эпитопов класса II. Приведенные ниже определения дополняют определения в данной области и относятся к настоящей заявке, и они не должны приписываться какому-либо имеющему или не имеющему отношению случаю, например, какому-либо патенту или заявке того же заявителя. Хотя для применения на практике для тестирования настоящего изобретения можно использовать способы и материалы, сходные или эквивалентные способам и материалам, описанным в настоящем описании, иллюстративные материалы и способы описаны в настоящем описании. Таким образом, терминология, используемая в настоящем описании, предназначена только для описания конкретных вариантов осуществления и не предназначена для ограничения.
[0474] В настоящем описании описаны способы получения персонализированной вакцины против злокачественной опухоли. Способ получения персонализированной вакцины против злокачественной опухоли может включать идентификацию пептидных последовательностей с мутацией, экспрессируемой в злокачественных клетках индивидуума; ввод информации об аминокислотных положениях идентифицированных пептидных последовательностей с использованием компьютерного процессора в машинно-обучаемую модель для прогнозирования презентации пептидов HLA с получением набора прогнозов презентации для идентифицированных пептидных последовательностей, причем каждый прогноз презентации отражает вероятность того, что один или более белков, кодируемых аллелем MHC класса II злокачественной клетки индивидуума будет презентировать данную последовательность идентифицированной пептидной последовательности; и выбор подгруппы пептидных последовательностей, идентифицированных на основе набора прогнозов презентации, для получения персонализированной вакцины против злокачественной опухоли.
[0475] В некоторых вариантах осуществления один или более результатов, полученных посредством способа, описанного в настоящем описании, могут обеспечить количественную величину или величины, указывающие на одно или более из следующих: вероятность диагностической точности, вероятность наличия состояния у индивидуума, вероятность развития у индивидуума состояния, вероятность успеха конкретного способа лечения или их комбинация. В некоторых вариантах осуществления способ, как описано в настоящем описании, может прогнозировать риск или вероятность развития состояния. В некоторых вариантах осуществления способ, как описано в настоящем описании, может предоставлять собой ранний диагностический индикатор развития состояния. В некоторых вариантах осуществления способ, как описано в настоящем описании, может подтвердить диагноз или наличие состояния. В некоторых вариантах осуществления способ, как описано в настоящем описании, может осуществлять мониторинг прогрессирования состояния. В некоторых вариантах осуществления способ, как описано в настоящем описании, может осуществлять мониторинг эффективности лечения состояния у индивидуума.
Способ идентификации пептидов MHC-II
[0476] В одном аспекте в рамках настоящего изобретения описан способ идентификации одного или более пептидов, которые презентируются белками MHC-II, для активации иммунитета. В некоторых вариантах осуществления один или более пептидов содержат эпитоп. В некоторых вариантах осуществления способ вовлекает компьютерное прогнозирование вероятности того, что конкретные эпитопы презентируются белком MHC-II. В некоторых вариантах осуществления способ вовлекает компьютерное прогнозирование специфичности эпитопа для презентации MHC-II. В некоторых вариантах осуществления компьютерные способы прогнозирования вовлекают оценку взаимодействий пептид-MHC. В некоторых вариантах осуществления компьютерные способы прогнозирования вовлекают прогнозирование аллельной специфичности пептида для презентации антигена. В некоторых вариантах осуществления компьютерные способы прогнозирования вовлекают интеграцию биоинформатической информации, например, нуклеотидных последовательностей, структурных мотивов биомолекул, признаков белок-белкового взаимодействия и функциональной эффективности, такой как иммуногенность. В некоторых вариантах осуществления компьютерные способы прогнозирования вовлекают машинное обучение. Было разработано много иммуноинформатических способов для прогнозирования взаимодействий пептид-MHC для MHC как класса I, так и класса II, на основе подходов машинного обучения, таких как мотив простого паттерна, метод опорных векторов (SVM), скрытая модель Маркова (HMM), модели нейронных сетей (NN), количественный анализ взаимосвязи структура-активность (QSAR), методы на основе структуры и биофизические способы. Эти способы могут быть подразделены на две категории, а именно, интрааллельные (аллелеспецифические) и трансаллельные (панспецифические) способы. Интрааллельные способы обучают для конкретной молекулы MHC на ограниченном наборе экспериментальных данных связывания пептидов, и их используют для прогнозирования пептидов, связывающихся с этой молекулой. Вследствие чрезвычайного полиморфизма молекул MHC, существования тысяч аллельных вариантов в комбинации с отсутствием достаточных экспериментальных данных связывания, является невозможным построить прогностическую модель для каждого аллеля. Таким образом, были разработаны трансаллельные способы и способы общего назначения, такие как NetMHCIIpan (Karosiene E etal., NetMHCIIpan-3.0, a common pan-specific MHC class II prediction method including all three human MHC class II isotypes, HLA-DR, HLA-DP and HLADQ. Immunogenetics (2013) 65(10):711-24), и TEPITOPEpan (Zhang L., et al., TEPITOPEpan: extending TEPITOPE for peptide binding prediction covering over 700 HLA-DR molecules. PLoS One (2012) 7(2):e30483) с использованием данных связывания пептидов, распространяющихся на множество аллелей или между видами. Также доступны сходные способы для MHC-I, такие как NetMHCpan и KISS.
[0477] В некоторых вариантах осуществления пептидные последовательности могут не экспрессироваться в нормальных клетках индивидуума. В некоторых вариантах осуществления любая клетка индивидуума может не быть злокачественной клеткой. Злокачественные клетки могут продуцироваться различными злокачественными опухолями, включая, но не ограничиваясь ими, рак щитовидной железы, рак коры надпочечников, рак анального канала, апластическую анемию, рак желчных протоков, рак мочевого пузыря, рак кости, метастазы в кость, злокачественные опухоли центральной нервной системы (ЦНС), злокачественные опухоли периферической нервной системы (PNS), рак молочной железы, болезнь Кастлмана, рак шейки матки, детскую неходжскинскую лимфому, лимфому, рак ободочной и прямой кишки, рак эндометрия, рак пищевода, семейство опухолей Юинга (например, саркома Юинга), рак глаза, рак желчного пузыря, желудочно-кишечные карциноидные опухоли, желудочно-кишечные стромальные опухоли, гестационную трофобластическую болезнь, волосатоклеточный лейкоз, болезнь Ходжкина, саркому Капоши, рак почки, рак гортани и гипофарингеальный рак, острый лимфоцитарный лейкоз, острый миелоидный лейкоз, детский лейкоз, хронический лимфоцитарный лейкоз, хронический миелоидный лейкоз, рак печени, рак легкого, карциноидные опухоли легкого, неходжскинскую лимфому, мужской рак молочной железы, злокачественную мезотелиому, множественную миелому, миелодиспластический синдром, миелопролиферативные нарушения, рак носовой полости и околоносовой рак, рак носоглотки, нейробластому, рак полости рта и ротоглотки, остеосаркому, рак яичника, рак поджелудочной железы, рак полового члена, опухоль гипофиза, рак предстательной железы, ретинобластому, рабдомиосаркому, рак слюнных желез, саркому (взрослый рак мягких тканей), меланомный рак кожи, немеланомный рак кожи, рак желудка, рак яичка, рак тимуса, рак тела матки (например, саркома матки), рак влагалища, рак вульвы или макроглобулинемию Вальденстрема.
[0478] Идентификация может включать сравнение последовательностей ДНК, РНК или белков из злокачественных клеток индивидуума с последовательностями ДНК, РНК или белков из нормальных клеток индивидуума. Последовательности ДНК, РНК или белов из злокачественных клеток индивидуума могут отличаться от последовательностей ДНК, РНК или белков нормальных клеток индивидуумов. Идентификация может идентифицировать варианты нуклеиновых кислот с высокой чувствительностью.
[0479] Машинно-обучаемая модель для прогнозирования презентации пептидов HLA может включать множество прогностических переменных, идентифицированных по меньшей мере на основе обучающих данных. Обучающие данные могут включать информацию о последовательности пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; обучающую информацию о пептидных последовательностях, включающую информацию об аминокислотных положениях, где обучающая информация о пептидных последовательностях ассоциирована с белком HLA, экспрессируемым в клетках; и функцию, отражающую взаимосвязь между информацией об аминокислотных положениях, введенной в качестве входных данных, и вероятностью презентации, сгенерированную в качестве результата на основе информации об аминокислотных положениях и прогностических переменных.
[0480] В некоторых вариантах осуществления обучающие данные, кроме того, могут включать структурированные данные, данные временного ряда, неструктурированные данные и реляционные данные. Неструктурированные данные могут включать аудиоданные, данные в виде изображений, видео, механические данные, электрические данные, химические данные и любую их комбинацию, для применения в точной имитации или обучении робототехники или имитационных моделей. Данные временного ряда могут включать данные от одного или более из интеллектуального счетчика, интеллектуального приспособления, интеллектуального устройства, системы мониторинга, устройства телеметрии или датчика. Реляционные данные включают данные системы потребителя, системы масштаба предприятия, операционной системы, веб-сайта, доступного через сеть прикладного программного интерфейса (API) или любой их комбинации. Введение данных может осуществлять пользователь посредством любого способа ввода файлов или других форматов данных в программное обеспечение или системы.
[0481] В некоторых вариантах осуществления обучающие данные могут храниться в базе данных. База данных может храниться в машиночитаемом формате. Компьютерный процессор может быть организован для доступа к данным, хранящимся в машиночитаемой памяти. В некоторых вариантах осуществления компьютерную систему можно использовать для анализа данных для получения результата. Результат может храниться удаленно или внутри носителя памяти и сообщаться персоналу, такому как медицинские специалисты. В некоторых вариантах осуществления компьютерная система может быть функционально связана с компонентами для передачи результата. Компоненты для передачи могут включать проводные и беспроводные компоненты. Примеры проводных коммуникационных компонентов могут включать соединение посредством универсальной последовательной шины (USB), соединение через коаксильный кабель, кабель Ethernet, такой как Cat5 или Cat6, оптоволоконный кабель или телефонную линию. Примеры беспроводных коммуникационных компонентов могут включать Wi-Fi-приемник, компонент для доступа к стандарту мобильной передачи данных, такому как сигнал данных 3G или 4G LTE, или Bluetooth-приемник. В некоторых вариантах осуществления все эти данные в носителе для хранения получаются и хранятся для построения хранилища данных.
[0482] В некоторых вариантах осуществления база данных включает внешнюю базу данных. Внешняя база данных может представлять собой медицинскую базу данных, например, но не ограничиваясь ими, Adverse Drug Effects Database, AHFS Supplemental File, Allergen Picklist File, Average WAC Pricing File, Brand Probability File, Canadian Drug File v2, Comprehensive Price History, Controlled Substances File, Drug Allergy Cross-Reference File, Drug Application File, Drug Dosing & Administration Database, Drug Image Database v2.0/Drug Imprint Database v2.0, Drug Inactive Date File, Drug Indications Database, Drug Lab Conflict Database, Drug Therapy Monitoring System (DTMS) v2.2/DTMS Consumer Monographs, Duplicate Therapy Database, Federal Government Pricing File, Healthcare Common Procedure Coding System Codes (HCPCS) Database, ICD-10 Mapping Files, Immunization Cross-Reference File, Integrated A to Z Drug Facts Module, Integrated Patient Education, Master Parameters Database, Medi-Span Electronic Drug File (MED-File) v2, Medicaid Rebate File, Medicare Plans File, Medical Condition Picklist File, Medical Conditions Master Database, Medication Order Management Database (MOMD), Parameters to Monitor Database, Patient Safety Programs File, Payment Allowance Limit-Part B (PAL-B) v2.0, Precautions Database, RxNorm Cross-Reference File, Standard Drug Identifiers Database, Substitution Groups File, Supplemental Names File, Uniform System of Classification Cross-Reference File или Warning Label Database.
[0483] В некоторых вариантах осуществления обучающие данные также могут быть получены посредством других источников данных. Источники данных могут включать датчики или интеллектуальные устройства, такие как приспособления, интеллектуальные счетчики, портативные электронные устройства, системы мониторинга, хранилища данных, системы потребителя, системы учета, финансовые системы, данные краудсорсинга, данные о погоде, социальные сети или любой другой датчик, систему масштаба предприятия или хранилище данных. Пример интеллектуальных счетчиков или датчиков может включать счетчики или датчики, находящиеся на территории потребителя, датчики или счетчики, находящиеся между потребителями и местом получения или источником. Путем включения данных из широкого набора источников, система может быть способна проводить комплексный и детальный анализ. В некоторых вариантах осуществления источники данных могут включать датчики или базы данных для других медицинских платформ без ограничений.
[0484] HLA-типирование обычно проводят либо посредством серологических способов с использованием антител, либо посредством способов на основе ПЦР, таких как последовательность-специфическая гибридизация олигонуклеотидных зондов (SSOP) или типирование на основе последовательности (SBT). В то время как первой из них препятствует потенциально высокая степень перекрестной реактивности и ограниченная разрешающая способность, вторую затрудняют проблемы, ассоциированные с эффективностью ПЦР вследствие в высокой степени ограниченных возможностей размещения праймеров вследствие полиморфных положений.
[0485] В некоторых вариантах осуществления информацию о последовательности идентифицируют либо способами секвенирования, либо способами с использованием масс-спектрометрии, такими как жидкостная хроматография-масс-спектрометрия (LC-MS или LC-MS/MS, или альтернативно ВЭЖХ-MS или ВЭЖХ-MS/MS). Эти способы секвенирования могут быть хорошо известны специалисту в данной области и рассмотрены в Medzihradszky KF and Chalkley RJ. Mass Spectrom Rev. 2015 Jan-Feb; 34(1):43-63. В некоторых вариантах осуществления масс-спектрометрия представляет собой моноаллельную масс-спектрометрию. В некоторых вариантах осуществления масс-спектрометрия может представлять собой анализ посредством MS, анализ посредством MS/MS, анализ посредством LC-MS/MS или их комбинацию. В некоторых вариантах осуществления анализ посредством MS может использоваться для определения массы интактного пептида. Например, определение может включать определение массы интактного пептида (например, анализ посредством MS). В некоторых вариантах осуществления анализ посредством MS/MS может использоваться для определения массы пептидных фрагментов. Например, определение может включать определение массы пептидных фрагментов, которое может использоваться для определения аминокислотной последовательности пептида или его части (например, анализ посредством MS/MS). В некоторых вариантах осуществления массу пептидных фрагментов можно использовать для определения последовательности аминокислот в пептиде. В некоторых вариантах осуществления анализ посредством LC-MS/MS может использовать для разделения комплексных пептидных смесей. Например, определение может включать разделение комплексных пептидных смесей, например, посредством жидкостной хроматографии, и определение массы интактного пептида, массы пептидных фрагментов или их комбинации (например, анализ посредством LC-MS/MS). Эти данные могут использоваться, например, для секвенирования пептидов.
[0486] В некоторых вариантах осуществления обучающая информация о пептидных последовательностях включает информацию об аминокислотных положениях обучающих пептидов. В некоторых вариантах осуществления обучающая информация о пептидных последовательностях включает не более чем приблизительно 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10% или менее информации о последовательностях пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии. В некоторых вариантах осуществления обучающая информация о пептидных последовательностях может включать по меньшей мере приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или более информации о последовательностях пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии.
[0487] Любая информация и данные могут быть в паре с индивидуумом, который является источником информации и данных. Индивидуум или медицинский специалист может извлекать информацию и данные из хранилища или сервера через идентификационную информацию индивидуума. Идентификационная информация индивидуума может включать фото пациента, имя, адрес, номер карточки социального обеспечения, дату рождения, телефонный номер, почтовый индекс или любую их комбинацию. Идентификационная информация индивидуума может быть зашифрована и закодирована в виде визуального графического кода. Визуальный графический код может представлять собой одноразовый штрихкод, который может быть уникально ассоциирован с индивидуальной информацией индивидуума. Штрихкод может представлять собой штрихкод UPC, штрихкод EAN, штрихкод Code 39, штрихкод Code 128, штрихкод ITF, штрихкод CodaBar, штрихкод GS1 DataBar, штрихкод MSI Plessey, штрихкод QR, код Datamatrix, код PDF417 или штрихкод Aztec. Визуальный графический код может быть организован для вывода на экран. Штрихкод может включать QR, который может быть оптически захвачен и считан устройством. Штрихкод может определять элемент, такой как версия, формат, положение, калибровку или время штрихкод, чтобы позволить считать и декодировать штрихкод. Штрихкод может кодировать различные типы информации в любом подходящем формате, такие как бинарная или буквенно-цифровая информация. QR-код может иметь различные размеры символов при условии, что QR-код может сканироваться с приемлемого расстояния визуализирующим устройством. QR-код может иметь любой формат файла изображения (например, векторные графики EPS или SVG, растровый графический формат PNG, TIF, GIF или JPEG).
[0488] В некоторых вариантах осуществления функция, отражающая взаимосвязь между информацией об аминокислотных положениях, введенной в качестве входной информации, и вероятностью презентации, сгенерированная в качестве результата на основе информации об аминокислотных положениях и прогностических переменных, включает линейную или нелинейную функцию. Функция может представлять собой, например, функцию активации блока линейной ректификации (ReLU), функцию активации Leaky ReLu, или другую функцию, такую как насыщающий гиперболический тангенс, идентичность, бинарный шаг, логистическая функция, arcTan, Softsign, параметрический блок линейной ректификации, экспоненциальный линейный элемент, softPlus, Bent identity, softExponential, синусоидную, Sinc, гауссова или сигмоидальная функция, или любая их комбинацию.
[0489] В некоторых вариантах осуществления линейную функцию получают посредством линейной регрессии. В некоторых вариантах осуществления линейная регрессия представляет собой способ прогнозирования целевой переменной посредством аппроксимации к наилучшей линейной зависимости между зависимой и независимой переменными. Наилучшее соответствие может означать, что сумма всех расстояний между кривой и фактическими наблюдениями в каждой точке является наименьшей. Линейная регрессия может включать простую линейную регрессию или множественную линейную регрессию. В простой линейной регрессии может использоваться одна независимая переменная для прогнозирования зависимой переменной. В множественных линейных регрессиях может использоваться более одной независимой переменной для прогнозирования зависимой переменной посредством аппроксимации к наилучший линейной зависимости. Нелинейная функция может быть получена посредством нелинейной регрессии. Нелинейная регрессия может представлять собой форму регрессионного анализа, в котором данные наблюдений моделируются посредством функции, которая является нелинейной комбинацией параметров модели и зависит от одной или более независимых переменных. Нелинейная регрессия может включать ступенчатую функцию, кусочную функцию, сплайн и обобщенную аддитивную модель.
[0490] В некоторых вариантах осуществления возможность презентации указывается посредством одномерных величин (например, вероятностей). В некоторых вариантах осуществления вероятность имеет конфигурацию, предназначенную для определения возможности того, что событие произойдет. В некоторых вариантах осуществления вероятность находится в диапазоне приблизительно от 0 до 1, от 0,1 до 0,9, от 0,2 до 0,8, от 0,3 до 0,7, или от 0,4 до 0,6. Чем выше вероятность события, тем более вероятно, что событие может произойти. В некоторых вариантах осуществления событие включает любой тип ситуации, включая в качестве неограничивающих примеров, то, будет ли HLA-пептид презентировать некоторый пептид с определенной информацией об аминокислотных положениях, и заболеет ли индивидуум, исходя из информации об аминокислотных положениях. В некоторых вариантах осуществления возможность может быть указана посредством многомерных величин. Многомерные величины могут быть предоставлены посредством многомерного пространства, тепловой карты или электронной таблицы.
[0491] В одном варианте осуществления выбор подгруппы пептидных последовательностей, идентифицированных на основе набора прогнозов презентации, направлен на получение персонализированной вакцины против злокачественной опухоли. В некоторых вариантах осуществления подгруппа включает не более чем приблизительно 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10% или менее пептидных последовательностей, идентифицированных на основе набора прогнозов презентации. В других случаях подгруппа может включать по меньшей мере приблизительно 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или более пептидных последовательностей, идентифицированных на основе набора прогнозов презентации. Вакцина против злокачественной опухоли может представлять собой вакцину, которая либо осуществляет лечение существующей злокачественной опухоли, либо препятствует развитию злокачественной опухоли. Вакцины можно получать из образцов, полученных от пациента, и они могут быть специфическими для этого пациента.
[0492] В некоторых вариантах осуществления в вакцине против заболевания (например, злокачественной опухоли) или иммуногенной композиции используются поксвирусы. Они включают ортопоксвирус, авипокс, вирус коровьей оспы, MVA, NYVAC, вирус оспы канареек, ALVAC, вирус оспы кур, TROVAC и т.д. Преимущества векторов могут включать простоту конструкции, возможность вмещать большие количества чужеродной ДНК и высокие уровни экспрессии. Информация, касающаяся поксвирусов, которые могут использоваться для применения настоящего изобретения на практике, таких как поксвирусы подсемейства Chordopoxvirinae (поксвирусы позвоночных), например, ортопоксвирусы и авипоксвирусы, например, вирус коровьей оспы (например, штамм Wyeth, штамм WR (например, ATCC® VR-1354), копенгагенский штамм, NYVAC, NYVAC.1, NYVAC.2, MVA, MVA-BN), вирус оспы канареек (например, штамм Wheatley C93, ALVAC), вирус оспы кур (например, штамм FP9, штамм Webster, TROVAC), вирус оспы горлиц, вирус оспы голубей, вирус оспы перепелок и вирус оспы енотов, среди прочих, их синтетических или не встречающихся в природе рекомбинантных форм, их применений и способов получения и применения таких рекомбинантных форм, могут быть найдены в научной и патентной литературе.
[0493] В некоторых вариантах осуществления в вакцине против заболевания или иммуногенной композиции для экспрессии антигена используют вирус коровьей оспы. Рекомбинантный вирус коровьей оспы может быть способен реплицироваться в цитоплазме инфицированной клетки-хозяина и представляющий интерес полипептид, таким образом, может индуцировать иммунный ответ.
[0494] В некоторых вариантах осуществления ALVAC используется в качестве вектора в вакцине против заболевания или иммуногенной композиции. ALVAC может представлять собой вирус оспы канареек, который может быть модифицирован для экспрессии чужеродных трансгенов и используется в качестве средства для вакцинации против как прокариотических, так и эукариотических антигенов.
[0495] В некоторых вариантах осуществления модифицированный вирус осповакцины Анкара (MVA) используют в качестве вирусного вектора для антигенной вакцины или иммуногенной композиции. MVA может быть представителем семейства ортопоксвирусов, и он получен посредством приблизительно 570 последовательных пассирований на фибробластах куриного эмбриона вируса коровьей оспы штамма Анкара (CVA). Вследствие этих пассирований полученный вирус MVA может включать на 31 тысячу пар оснований меньше информации по сравнению с CVA, и он является в высокой степени ограниченным в отношении клеток-хозяев. MVA может характеризоваться его чрезвычайной аттенуацией, а именно, сниженной вирулентностью или инфекционной способностью, однако он все еще сохраняет превосходную иммуногенность. При тестировании в различных моделях на животных может быть показано, что MVA является авирулентным даже у индивидуумов с иммуносупрессией. Более того, MVA-BN®-HER2 может быть иммунотерапевтическим средством-кандидатом, предназначенным для лечения положительного по HER-2 рака молочной железы, и в настоящее время он проходит клинические испытания.
[0496] В некоторых вариантах осуществления в качестве части модели прогнозирования используют положительную прогностическую величину (PPV). PPV, также известная как показатель точности, представляет собой вероятность того, что индивидуум, у которого диагностировано заболевание или состояние посредством, например, теста или модели, в действительности имеет заболевание или состояние. Ее можно вычислять путем деления количества истинных положительных результатов на общее количество результатов, которые выданы в качестве положительных (результаты, которые включают ложноположительные результаты). PPV = истинные положительные результаты / (истинные положительные результаты + ложноположительные результаты). Например, если в выборке из 100 пациентов модель идентифицировала положительный результат у 50 пациентов, среди которых 25 были истинно положительными, PPV будет составлять 25/50=0,5. PPV, близкая к 1, отражает более точный способ диагностики, такой как тест или модель. PPV может использоваться для определения точности прогностической модели. PPV может использоваться для коррекции прогностической модели для учета ложноположительных результатов, которые могут быть получены посредством данной модели.
[0497] В качестве части прогностической модели может использоваться частота отзыва. Частотой отзыва можно считать процент истинных положительных результатов из общего количества положительных результатов в выборке. Отзыв = истинно положительные результаты / (истинно положительные результаты + ложноотрицательные результаты). Например, если в выборке из 100 пациентов модель идентифицировала положительный результат у 50 пациентов, из которых 25 были истинно положительными и всего в выборке пациентов было 75 положительных результатов, частота отзыва будет {25/(25+25)} × 100=50%. Частота отзыва может использоваться для определения точности прогностической модели. Частота отзыва может использоваться для коррекции прогностической модели для учета ложноположительных результатов или ложноотрицательных результатов, которые могут быть получены посредством модели.
[0498] В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва 0,1%-10%. В некоторых вариантах осуществления прогностическая модель может иметь положительную прогностическую величину не более 0,9, 0,8, 0,7, 0,6, 0,5, 0,4, 0,3, 0,2, 0,1 или менее при частоте отзыва 0,1%-10%. Прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва менее 0,1%. В некоторых вариантах осуществления прогностическая модель может иметь положительную прогностическую величину не более 0,9, 0,8, 0,7, 0,6, 0,5, 0,4, 0,3, 0,2, 0,1 или менее при частоте отзыва менее 0,1%. Прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва более 10%. В некоторых вариантах осуществления прогностическая модель может иметь положительную прогностическую величина не более 0,9, 0,8, 0,7, 0,6, 0,5, 0,4, 0,3, 0,2, 0,1 или менее при частоте отзыва более 10%.
[0499] В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва от 0,1% до 10%. В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва от 0,1% до 0,5%, от 0,1% до 1%, от 0,1% до 2%, от 0,1% до 3%, от 0,1% до 4%, от 0,1% до 5%, от 0,1% до 6%, от 0,1% до 7%, от 0,1% до 8%, от 0,1% до 9%, от 0,1% до 10%, от 0,5% до 1%, от 0,5% до 2%, от 0,5% до 3%, от 0,5% до 4%, от 0,5% до 5%, от 0,5% до 6%, от 0,5% до 7%, от 0,5% до 8%, от 0,5% до 9%, от 0,5% до 10%, от 1% до 2%, от 1% до 3%, от 1% до 4%, от 1% до 5%, от 1% до 6%, от 1% до 7%, от 1% до 8%, от 1% до 9%, от 1% до 10%, от 2% до 3%, от 2% до 4%, от 2% до 5%, от 2% до 6%, от 2% до 7%, от 2% до 8%, от 2% до 9%, от 2% до 10%, от 3% до 4%, от 3% до 5%, от 3% до 6%, от 3% до 7%, от 3% до 8%, от 3% до 9%, от 3% до 10%, от 4% до 5%, от 4% до 6%, от 4% до 7%, от 4% до 8%, от 4% до 9%, от 4% до 10%, от 5% до 6%, от 5% до 7%, от 5% до 8%, от 5% до 9%, от 5% до 10%, от 6% до 7%, от 6% до 8%, от 6% до 9%, от 6% до 10%, от 7% до 8%, от 7% до 9%, от 7% до 10%, от 8% до 9%, от 8% до 10% или от 9% до 10%. В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9% или 10%. В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва по меньшей мере 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8% или 9%. В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва не более 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9% или 10%.
[0500] В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва от 10% до 20%. В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва от 10% до 11%, от 10% до 12%, от 10% до 13%, от 10% до 14%, от 10% до 15%, от 10% до 16%, от 10% до 17%, от 10% до 18%, от 10% до 19%, от 10% до 20%, от 11% до 12%, от 11% до 13%, от 11% до 14%, от 11% до 15%, от 11% до 16%, от 11% до 17%, от 11% до 18%, от 11% до 19%, от 11% до 20%, от 12% до 13%, от 12% до 14%, от 12% до 15%, от 12% до 16%, от 12% до 17%, от 12% до 18%, от 12% до 19%, от 12% до 20%, от 13% до 14%, от 13% до 15%, от 13% до 16%, от 13% до 17%, от 13% до 18%, от 13% до 19%, от 13% до 20%, от 14% до 15%, от 14% до 16%, от 14% до 17%, от 14% до 18%, от 14% до 19%, от 14% до 20%, от 15% до 16%, от 15% до 17%, от 15% до 18%, от 15% до 19%, от 15% до 20%, от 16% до 17%, от 16% до 18%, от 16% до 19%, от 16% до 20%, от 17% до 18%, от 17% до 19%, от 17% до 20%, от 18% до 19%, от 18% до 20%, или от 19% до 20%. В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20%. В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва по меньшей мере 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18% или 19%. В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва не более 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20%.
[0501] В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва по меньшей мере 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва по меньшей мере 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,2 при частоте отзыва по меньшей мере 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,3 при частоте отзыва по меньшей мере 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,4 при частоте отзыва по меньшей мере 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,5 при частоте отзыва по меньшей мере 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,6 при частоте отзыва по меньшей мере 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,7 при частоте отзыва по меньшей мере 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,8 при частоте отзыва по меньшей мере 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,9 при частоте отзыва по меньшей мере 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва по меньшей мере 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,2 при частоте отзыва по меньшей мере 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,3 при частоте отзыва по меньшей мере 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,4 при частоте отзыва по меньшей мере 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,5 при частоте отзыва по меньшей мере 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,6 при частоте отзыва по меньшей мере 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,7 при частоте отзыва по меньшей мере 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,8 при частоте отзыва по меньшей мере 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,9 при частоте отзыва по меньшей мере 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва по меньшей мере 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,2 при частоте отзыва по меньшей мере 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,3 при частоте отзыва по меньшей мере 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,4 при частоте отзыва по меньшей мере 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,5 при частоте отзыва по меньшей мере 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,6 при частоте отзыва по меньшей мере 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,7 при частоте отзыва по меньшей мере 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,8 при частоте отзыва по меньшей мере 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,9 при частоте отзыва по меньшей мере 20%.
[0502] В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва приблизительно 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва приблизительно 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,2 при частоте отзыва приблизительно 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,3 при частоте отзыва приблизительно 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,4 при частоте отзыва приблизительно 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,5 при частоте отзыва приблизительно 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,6 при частоте отзыва приблизительно 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,7 при частоте отзыва приблизительно 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,8 при частоте отзыва приблизительно 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,9 при частоте отзыва приблизительно 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва приблизительно 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,2 при частоте отзыва приблизительно 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,3 при частоте отзыва приблизительно 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,4 при частоте отзыва приблизительно 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,5 при частоте отзыва приблизительно 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,6 при частоте отзыва приблизительно 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,7 при частоте отзыва приблизительно 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,8 при частоте отзыва приблизительно 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,9 при частоте отзыва приблизительно 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва приблизительно 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,2 при частоте отзыва приблизительно 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,3 при частоте отзыва приблизительно 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,4 при частоте отзыва приблизительно 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,5 при частоте отзыва приблизительно 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,6 при частоте отзыва приблизительно 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,7 при частоте отзыва приблизительно 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,8 при частоте отзыва приблизительно 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,9 при частоте отзыва приблизительно 20%.
[0503] В некоторых вариантах осуществления прогностическая модель имеет положительную прогностическую величину по меньшей мере 0,05, 0,1, 0,2, 0,25, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9 или более при частоте отзыва менее 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва не более 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,2 при частоте отзыва не более 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,3 при частоте отзыва не более 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,4 при частоте отзыва не более 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,5 при частоте отзыва не более 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,6 при частоте отзыва не более 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,7 при частоте отзыва не более 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,8 при частоте отзыва не более 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,9 при частоте отзыва не более 10%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва не более 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,2 при частоте отзыва не более 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,3 при частоте отзыва не более 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,4 при частоте отзыва не более 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,5 при частоте отзыва не более 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,6 при частоте отзыва не более 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,7 при частоте отзыва не более 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,8 при частоте отзыва не более 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,9 при частоте отзыва не более 5%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,1 при частоте отзыва не более 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,2 при частоте отзыва не более 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,3 при частоте отзыва не более 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,4 при частоте отзыва не более 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,5 при частоте отзыва не более 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,6 при частоте отзыва не более 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,7 при частоте отзыва не более 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,8 при частоте отзыва не более 20%. Например, прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,9 при частоте отзыва не более 20%.
[0504] В некоторых вариантах осуществления при частоте отзыва приблизительно 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20% прогностическая модель имеет положительную прогностическую величину от 0,05% до 0,6%. При частоте отзыва приблизительно 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20% прогностическая модель может иметь положительную прогностическую величину от 0,05% до 0,1%, от 0,05% до 0,15%, от 0,05% до 0,2%, от 0,05% до 0,25%, от 0,05% до 0,3%, от 0,05% до 0,35%, от 0,05% до 0,4%, от 0,05% до 0,45%, от 0,05% до 0,5%, от 0,05% до 0,55%, от 0,05% до 0,6%, от 0,1% до 0,15%, от 0,1% до 0,2%, от 0,1% до 0,25%, от 0,1% до 0,3%, от 0,1% до 0,35%, от 0,1% до 0,4%, от 0,1% до 0,45%, от 0,1% до 0,5%, от 0,1% до 0,55%, от 0,1% до 0,6%, от 0,15% до 0,2%, от 0,15% до 0,25%, от 0,15% до 0,3%, от 0,15% до 0,35%, от 0,15% до 0,4%, от 0,15% до 0,45%, от 0,15% до 0,5%, от 0,15% до 0,55%, от 0,15% до 0,6%, от 0,2% до 0,25%, от 0,2% до 0,3%, от 0,2% до 0,35%, от 0,2% до 0,4%, от 0,2% до 0,45%, от 0,2% до 0,5%, от 0,2% до 0,55%, от 0,2% до 0,6%, от 0,25% до 0,3%, от 0,25% до 0,35%, от 0,25% до 0,4%, от 0,25% до 0,45%, от 0,25% до 0,5%, от 0,25% до 0,55%, от 0,25% до 0,6%, от 0,3% до 0,35%, от 0,3% до 0,4%, от 0,3% до 0,45%, от 0,3% до 0,5%, от 0,3% до 0,55%, от 0,3% до 0,6%, от 0,35% до 0,4%, от 0,35% до 0,45%, от 0,35% до 0,5%, от 0,35% до 0,55%, от 0,35% до 0,6%, от 0,4% до 0,45%, от 0,4% до 0,5%, от 0,4% до 0,55%, от 0,4% до 0,6%, от 0,45% до 0,5%, от 0,45% до 0,55%, от 0,45% до 0,6%, от 0,5% до 0,55%, от 0,5% до 0,6% или от 0,55% до 0,6%. При частоте отзыва приблизительно 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20% прогностическая модель может иметь положительную прогностическую величину 0,05%, 0,1%, 0,15%, 0,2%, 0,25%, 0,3%, 0,35%, 0,4%, 0,45%, 0,5%, 0,55% или 0,6%. При частоте отзыва приблизительно 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19%, или 20% прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,05%, 0,1%, 0,15%, 0,2%, 0,25%, 0,3%, 0,35%, 0,4%, 0,45%, 0,5% или 0,55%. При частоте отзыва приблизительно 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20% прогностическая модель может иметь положительную прогностическую величину не более 0,1%, 0,15%, 0,2%, 0,25%, 0,3%, 0,35%, 0,4%, 0,45%, 0,5%, 0,55% или 0,6%.
[0505] При частоте отзыва приблизительно 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20% прогностическая модель может иметь положительную прогностическую величину от 0,45% до 0,98%. При частоте отзыва приблизительно 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20% прогностическая модель может иметь положительную прогностическую величину от 0,45% до 0,5%, от 0,45% до 0,55%, от 0,45% до 0,6%, от 0,45% до 0,65%, от 0,45% до 0,7%, от 0,45% до 0,75%, от 0,45% до 0,8%, от 0,45% до 0,85%, от 0,45% до 0,9%, от 0,45% до 0,96%, от 0,45% до 0,98%, от 0,5% до 0,55%, от 0,5% до 0,6%, от 0,5% до 0,65%, от 0,5% до 0,7%, от 0,5% до 0,75%, от 0,5% до 0,8%, от 0,5% до 0,85%, от 0,5% до 0,9%, от 0,5% до 0,96%, от 0,5% до 0,98%, от 0,55% до 0,6%, от 0,55% до 0,65%, от 0,55% до 0,7%, от 0,55% до 0,75%, от 0,55% до 0,8%, от 0,55% до 0,85%, от 0,55% до 0,9%, от 0,55% до 0,96%, от 0,55% до 0,98%, от 0,6% до 0,65%, от 0,6% до 0,7%, от 0,6% до 0,75%, от 0,6% до 0,8%, от 0,6% до 0,85%, от 0,6% до 0,9%, от 0,6% до 0,96%, от 0,6% до 0,98%, от 0,65% до 0,7%, от 0,65% до 0,75%, от 0,65% до 0,8%, от 0,65% до 0,85%, от 0,65% до 0,9%, от 0,65% до 0,96%, от 0,65% до 0,98%, от 0,7% до 0,75%, от 0,7% до 0,8%, от 0,7% до 0,85%, от 0,7% до 0,9%, от 0,7% до 0,96%, от 0,7% до 0,98%, от 0,75% до 0,8%, от 0,75% до 0,85%, от 0,75% до 0,9%, от 0,75% до 0,96%, от 0,75% до 0,98%, от 0,8% до 0,85%, от 0,8% до 0,9%, от 0,8% до 0,96%, от 0,8% до 0,98%, от 0,85% до 0,9%, от 0,85% до 0,96%, от 0,85% до 0,98%, от 0,9% до 0,96%, от 0,9% до 0,98%, или от 0,96% до 0,98%. При частоте отзыва приблизительно 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20% прогностическая модель может иметь положительную прогностическую величину 0,45%, 0,5%, 0,55%, 0,6%, 0,65%, 0,7%, 0,75%, 0,8%, 0,85%, 0,9%, 0,96%, или 0,98%. При частоте отзыва приблизительно 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19%, или 20% прогностическая модель может иметь положительную прогностическую величину по меньшей мере 0,45%, 0,5%, 0,55%, 0,6%, 0,65%, 0,7%, 0,75%, 0,8%, 0,85%, 0,9%, или 0,96%. При частоте отзыва приблизительно 0,1%, 0,5%, 1%, 2%, 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15%, 16%, 17%, 18%, 19% или 20% прогностическая модель может иметь положительную прогностическую величину не более 0,5%, 0,55%, 0,6%, 0,65%, 0,7%, 0,75%, 0,8%, 0,85%, 0,9%, 0,96% или 0,98%.
Способы обучения машинно-обучаемой модели для прогнозирования презентации пептидов HLA
[0506] В одном аспекте способ обучения машинно-обучаемой модели для прогнозирования презентации пептидов HLA может включать ввод информации об аминокислотных положениях для последовательностей HLA-пептидов, выделенных из одного или более комплексов HLA-пептид в клетке, экспрессирующей аллель HLA класса II в модели прогнозирования презентации пептидов HLA с использованием компьютерного процессора; обучение машинно-обучаемой модели для прогнозирования презентации пептидов HLA может включать коррекцию взвешенных величин для узлов нейронной сети для наилучшего соответствия предоставленным обучающим данным.
[0507] Обучающие данные могут включать информацию о последовательностях пептидов, презентируемых белком HLA, экспрессируемым в клетках, и идентифицированных посредством масс-спектрометрии; обучающую информацию о пептидных последовательностях, включающую информацию об аминокислотных положениях обучающих пептидов, где обучающая информация о пептидных последовательностях ассоциирована с белком HLA, экспрессируемым в клетках; и функцию, отражающую взаимосвязь между информацией об аминокислотных положениях, полученной на входе, и вероятностью презентации, сгенерированной на выходе на основе информации об аминокислотных положениях и прогностических переменных. Обучающие данные, обучающая информация о пептидных последовательностях, функция и вероятность презентации описаны в настоящем описании.
[0508] Обученный алгоритм может включать одну или более нейронных сетей, нейронная сеть может представлять собой тип вычислительной системы на основе графа из нескольких соединенных нейронов (или узлов) в серии слоев. Нейронная сеть может включать входной слой, на который предоставляются данные; один или более внутренних и/или "скрытых" слоев; и выходной слой, из которого предоставляются результаты. Нейронная сеть может выучивать взаимосвязи между входным набором данных и целевым набором данных посредством установления серии весов связи. Нейрон может быть соединен с нейронами в других слоях посредством связей, которые имеют веса, которые являются параметрами, которые контролируют силу связи. Количество нейронов в каждом слое может быть связано со сложностью проблемы, подлежащей решению. Минимальное количество нейронов, требуемое в слое, может определяться сложностью проблемы, и максимальное количество может ограничиваться способностью нейронной сети к обобщению. Входные нейроны могут получать предоставляемые данные, а затем передавать эти данные на узел в первом скрытом слое через веса связей, которые модифицируются в ходе обучения. Узел результата может суммировать результаты произведения для всех пар вводных данных и ассоциированных с ними весов. Взвешенная сумма может быть компенсирована посредством смещения для внесения поправки на величину узла результата. Результат узла или нейрона может пропускаться с использованием пороговой или активационной функции. Активационная функция может представлять собой линейную или нелинейную функцию. Активационная функция может представлять собой, например, активационную функцию блока линейной ректификации (ReLU), активационную функцию Leaky ReLu или другую функцию, такую как насыщающий гиперболический тангенс, идентичность, бинарный шаг, логистическая функция, arcTan, Softsign, параметрический блок линейной ректификации, экспоненциальный линейный элемент, softPlus, Bent identity, softExponential, синусоидная, Sinc, гауссова или сигмоидальная функция или любая их комбинация.
[0509] Скрытый слой в нейронной сети может обрабатывать данные и передавать результат на следующий слой через группу взвешенных соединений. Каждый последующий слой может "объединять" результатов из предшествующих слоев в более комплексные взаимосвязи. Нейронные сети могут быть обучены с использованием известной выборки обучающих данных (данные, полученные от одного или более датчиков) путем позволения им модифицировать себя в процессе (и после) обучения для получения желаемого результата из данного набора входных данных, такого как выходная величина. Обученный алгоритм может включать сверточные нейронные сети, рекуррентные нейронные сети, расширенные сверточные нейронные сети, полносвязанные нейронные сети, глубокие генеративные модели и машины Больцмана.
[0510] Коэффициенты взвешивания, величины смещения и пороговые величины или другие вычислительные параметры нейронной сети, могут быть "выучены" или "изучены" в обучающей фазе с использованием одной или более выборок обучающих данных. Например, параметры могут быть выучены с использованием входных данных из обучающего набора данных и способа градиентного спуска или обратного распространения, так чтобы выходная величина(ы) из нейронной сети согласовывалась с примерами, включенными в обучающий набор данных.
[0511] Количество узлов, используемых во входном слое нейронной сети, может составлять по меньшей мере приблизительно 10, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 2000, 3000, 4000, 5000, 6000, 7000, 8000, 9000, 10000, 20000, 30000, 40000, 50000, 60000, 70000, 80000, 90000, 100000 или более. В других случаях количество узлов, используемых во входном слое, может составлять не более чем приблизительно 100000, 90000, 80000, 70000, 60000, 50000, 40000, 30000, 20000, 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000, 1000, 900, 800, 700, 600, 500, 400, 300, 200, 100, 50 или 10 или менее. В некоторых случаях количество слоев, используемых в нейронной сети (включая входной и выходной слои) может составлять по меньшей мере приблизительно 3, 4, 5, 10, 15, 20 или более. В других случаях общее количество слоев может составлять приблизительно 20, 15, 10, 5, 4, 3 или менее.
[0512] В некоторых случаях общее количество поддающихся обучению или тренировке параметров, например, коэффициенты взвешивания, смещения или пороговые величины, используемые в нейронной сети, могут составлять по меньшей мере приблизительно 10, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 2000, 3000, 4000, 5000, 6000, 7000, 8000, 9000, 10000, 20000, 30000, 40000, 50000, 60000, 70000, 80000, 90000, 100000 или более. В других случаях количество поддающихся обучению параметров может составлять не более чем приблизительно 100000, 90000, 80000, 70000, 60000, 50000, 40000, 30000, 20000, 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000, 1000, 900, 800, 700, 600, 500, 400, 300, 200, 100, 50 или 10 или менее.
[0513] Нейронная семь может включать сверточную нейронную сеть. Сверточная нейронная сеть может включать один или более сверточных слоев, расширенных слоев или полносвязных слоев. Количество сверточных слоев может составлять 1-10 и количество расширенных слоев может составлять 0-10. Общее количество сверточных слоев (включая входной и выходной слои) может составлять по меньшей мере приблизительно 1,2, 3, 4, 5, 10, 15, 20 или более, и общее количество расширенных слоев может составлять по меньшей мере приблизительно 1,2, 3, 4, 5, 10, 15, 20 или более. Общее количество сверточных слоев может составлять не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее, и общее количество расширенных слоев может составлять не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее. В некоторых вариантах осуществления количество сверточных слоев составляет 1-10 и количество полносвязных слоев составляет 0-10. Общее количество сверточных слоев (включая входной и выходной слои) может составлять по меньшей мере приблизительно 1,2, 3, 4, 5, 10, 15, 20 или более, и общее количество полносвязных слоев может составлять по меньшей мере приблизительно 1,2, 3, 4, 5, 10, 15, 20 или более. Общее количество сверточных слоев может составлять не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее, и обще количество полносвязных слоев может составлять не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее.
[0514] Сверточная нейронная сеть (CNN) может представлять собой глубокую и упреждающую искусственную нейронную сеть. CNN может использоваться для анализа зрительных образов. CNN может включать входной, выходной слой и множество скрытых слоев. Скрытые слои CNN могут включать сверточные слои, слои пулинга, полносвязные слои и слои нормализации. Слои могут быть организованы в 3 измерениях: по ширине, высоте и глубине.
[0515] Сверточные слои может применять операцию свертывания для ввода и передачи результатов операции свертывания на следующий слой. Для обработки изображений операция свертывания может уменьшать количество свободных параметров, позволяя сети быть глубже при меньшем количестве параметров. В сверточном слое нейроны могут принимать входную информацию только из ограниченной подобласти предыдущего слоя. Параметры сверточного слоя могут включать набор обучаемых фильтров (или ядер). Обучаемые фильтры могут иметь маленькое рецептивное поле и протягиваться на полную глубину объема ввода. В ходе прямого прохождения каждый фильтр может свертываться по ширине и высоте вводного объема, вычислять скалярное произведение между данными на фильтре и вводом, и генерировать 2-мерную активационную карту для этого фильтра. В результате сеть может выучивать фильтры, которые активируются, когда они выявляют некоторый конкретный тип признака в некотором пространственном положении на входе.
[0516] Слои пулинга могут включать слои глобального пулинга. Слои глобального пулинга могут объединять результаты нейронных кластеров одного слоя в один нейрон следующего слоя. Например, слои максимального пулинга могут использовать максимальную величину из каждого кластера нейронов на предыдущем слое; и слои усредненного пулинга могут использовать среднюю величину из каждого кластера нейронов на предыдущем слое. Полносвязные слои могут соединять каждый нейрон в одном слое с каждым нейроном в другом слое. В полносвязном слое каждый нейрон может получать входную информацию от каждого элемента предыдущего слоя. Слой нормализации может представлять собой слой пакетной нормализации. Слой пакетной нормализации может повышать эффективность и стабильность нейронных сетей. Слой пакетной нормализации может представлять собой любой слой нейронной сети с входными данными, которые имеют нулевую среднюю/единичную дисперсию. Преимущества использования слоя пакетной нормализации могут включать более быстро обучаемые сети, более высокие показатели обучения, упрощение инициализации весов, более эффективные активационные функции и более простой процесс создания глубоких сетей.
[0517] Нейронная сеть может включать рекуррентную нейронную сеть. Рекуррентная нейронная сеть может быть организована для получения секвенциальных данных на входе, таких как последовательные входные данные, и модуль программного обеспечения рекуррентной нервной сети может обновлять внутреннее состояние на каждом временном шагу. Рекуррентная нейронная сеть может использовать внутреннее состояние (память) для обработки последовательностей вводных данных. Рекуррентная нейронная сеть может быть применима к таким задачам, как распознавание почерка или распознавание речи, предсказание следующего слова, составление музыкального произведения, подписи к изображениям, обнаружение отклонений временных рядов, машинный перевод, сценическая маркировка и прогнозирование рынка ценных бумаг. Рекуррентная нейронная сеть может включать полностью рекуррентную нейронную сеть, независимо рекуррентную нейронную сеть, нейронную сеть Элмана, сети Джордана, сеть эхо-состояний, нейронный компрессор истории, долгую краткосрочную память, управляемый рекуррентный блок, модель множественных временных шкал, нейронные машины Тьюринга, дифференцируемый нейрокомпьютер, автоматы с магазинной памятью на основе нейронных сетей или любую их комбинацию.
[0518] Обученный алгоритм может включать способ обучения с учителем или без учителя, например, такой как SVM, случайные леса, алгоритм кластеризации (или модуль программного обеспечения), градиентный бустинг, логистическая регрессия и/или деревья решений. Алгоритмы обучения с учителем могут представлять собой алгоритмы, которые основаны на применении набора меченых парных обучающих примеров данных для установления взаимосвязи между входными данными и выходными данными. Алгоритмы обучения без учителя могут представлять собой алгоритмы, используемые для установления выводов от обучающих данных к выходным данным. Алгоритмы обучения без учителя могут включать кластерный анализ, который может использоваться для анализа данных исследований для поиска скрытых паттернов или групп в данных обработки. Один из примеров способа обучения без учителя может включать анализ главных компонентов. Анализ главных компонентов может включать уменьшение размерности одной или более переменных. Размерность данных переменных может составлять по меньшей мере 1, 5, 10, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1100, 1200 1300, 1400, 1500, 1600, 1700, 1800 или более. Размерность данных переменных может составлять не более 1800, 1600, 1500, 1400, 1300, 1200, 1100, 1000, 900, 800, 700, 600, 500, 400, 300, 200, 100, 50, 10 или менее.
[0519] Алгоритм обучения может быть получен посредством статистических способов. В некоторых вариантах осуществления статистические способы могут включать линейную регрессию, классификацию, способы перевыборки, выбор подмножества, стягивание, снижение размерности, нелинейные модели, способы на основе деревьев, метод опорных векторов, обучение без учителя или их комбинацию.
[0520] Линейная регрессия может представлять собой способ прогнозирования целевой переменной посредством аппроксимации к наилучший линейной зависимости зависимой и независимой переменных. Наилучшее соответствие может означать, что сумма всех расстояний между кривой и фактическими наблюдениями в каждой точке является наименьшей. Линейная регрессия может включать простую линейную регрессию или множественную линейную регрессию. В простой линейной регрессии может использоваться одна независимая переменная для прогнозирования зависимой переменной. В множественных линейных регрессиях может использоваться более одной независимой переменной для прогнозирования зависимой переменной посредством аппроксимации к наилучший линейной зависимости.
[0521] Классификация может представлять собой способ интеллектуального анализа данных, который присваивает категории набору данных для достижения точного прогнозирования и анализа. Способы классификации могут включать логистическую регрессию и дискриминационный анализ. Логистическая регрессия может использоваться, когда зависимая переменная является дихотомической (бинарной). Логистическая регрессия может использоваться для обнаружения и описания взаимосвязи между одной зависимой бинарной переменной и одной или более номинальными, порядковыми независимыми переменными, независимыми переменными на уровне интервалов или соотношений. Перевыборка может представлять собой способ, включающий отбор повторных выборок из первоначальных выборок данных. Перевыборка может не вовлекать использование общих таблиц распределения для вычисления приблизительных величин вероятности. Перевыборка может осуществлять уникальное выборочное распределение на основе фактических данных. В некоторых вариантах осуществления в перевыборке могут использоваться экспериментальные способы, а не аналитические способы, для получения уникального выборочного распределения. Способы перевыборки могут включать взятие бутстрап-выборок и перекрестную валидацию. Взятие бутстрап-выборок можно проводить посредством выборки с возвращением из исходных данных, и она принимает "не выбранные" точки данных в качестве тестовых случаев. Перекрестную валидацию можно проводить посредством разделения обучающих данных на множество частей.
[0522] Выбор подмножества может идентифицировать подмножество прогностических факторов, связанных с ответом. Выбор подмножества может включать выбор наилучшего подмножества, прямой ступенчатый выбор, обратный ступенчатый выбор, гибридный способ или любую их комбинацию. В некоторых вариантах осуществления стягивание соответствует модели, вовлекающей все прогностические факторы, однако оцененные коэффициенты сжимаются к нулю относительно оценок методом наименьших квадратов. Это стягивание может уменьшать дисперсию. Стягивание может включать гребневую регрессию и лассо. Снижение размерности может уменьшать проблему оценки коэффициентов n+1 до более простой проблемы коэффициентов m+1, где m<n. Оно может быть достигнуто путем вычисления n различных линейных комбинаций или проекций переменных. Затем эти n проекций используются в качестве прогностических параметров для аппроксимации линейной регрессионной модели посредством метода наименьших квадратов. Снижение размерности может включать регрессию по основным компонентам и частичные наименьшие квадраты. Регрессия по основным компонентам может использоваться для установления низкоразмерного набора признаков из большого набора переменных. Основной компонент, используемый в регрессии по основным компонентам, может выявлять наибольшую дисперсию данных с использованием линейных комбинаций данных в последовательно ортогональных направлениях. Частичные наименьшие квадраты могут быть альтернативой с учителем для регрессии по основным компонентам, поскольку в частичных наименьших квадратах может использовать переменная ответа для идентификации новых признаков.
[0523] Нелинейная регрессия может представлять собой форму регрессионного анализа, в котором данные наблюдений моделируются посредством функции, которая представляет собой нелинейную комбинацию модельных параметров и зависит от одной или более независимых переменных. Нелинейная регрессия может включать ступенчатую функцию, кусочную функцию, сплайн, генерализованную аддитивную модель или их комбинацию.
[0524] Способы на основе деревьев могут использоваться как для проблем регрессии, так и для проблем классификации. Проблемы регрессии и классификации могут вовлекать стратификацию или сегментацию прогностического пространства на ряд простых областей. Способы на основе деревьев могут включать создание подмножеств, бустинг, случайный лес или любую их комбинацию. Создание подмножеств может уменьшать дисперсию прогнозирования посредством создания дополнительных данных для обучения из первоначального набора данных с использованием комбинаций с повторами для получения многоступенчатых данных с тем же чувствительностью/размером, что и у исходных данных. Бустинг может вычислять результат с использованием нескольких различных моделей, а затем усреднять результат с использованием подхода средневзвешенных значений. Алгоритм случайного леса может осуществлять взятие случайных бутстрэп-выборок из обучающего набора. Метод опорных векторов может представлять собой способ классификации. Способ опорных векторов может включать поиск гиперплоскости, которая наилучшим образом разделяет два класса точек с максимальной разницей. Метод опорных векторов может минимизировать проблему оптимизации, так чтобы разница максимизировалась с учетом ограничения, которое безупречно классифицирует данные.
[0525] Способы без учителя могут представлять собой способы вывода заключений из наборов данных, включающих вводные данные без маркированных ответов. Способы без учителя могут включать кластеризацию, анализ главных компонентов, кластеризацию методом k-средних, иерархическую кластеризацию или любую их комбинацию.
[0526] Масс-спектрометрия может представлять собой моноаллельную масс-спектрометрию. В некоторых вариантах осуществления масс-спектрометрия может представлять собой анализ посредством MS, анализ посредством MS/MS, анализ посредством LC-MS/MS или их комбинацию. В некоторых вариантах осуществления анализ посредством MS может использоваться для определения массы интактного пептида. Например, определение может включать определение массы интактного пептида (например, анализ MS). В некоторых вариантах осуществления анализ посредством MS/MS может использоваться для определения массы пептидных фрагментов. Например, определение может включать определение массы пептидных фрагментов, которые могут использоваться для определения аминокислотной последовательности пептида или его части (например, анализ посредством MS/MS). В некоторых вариантах осуществления масса пептидных фрагментов может использоваться для определения последовательности аминокислот в пептиде. В некоторых вариантах осуществления анализ посредством LC-MS/MS может использоваться для разделения комплексных пептидных смесей. Например, определение может включать разделение комплексных пептидных смесей, например, посредством жидкостной хроматографии, и определение массы интактного пептида, массы пептидных фрагментов или их комбинации (например, анализ посредством LC-MS/MS). Эти данные могут использоваться, например, для секвенирования пептидов.
[0527] Пептиды могут презентироваться белком HLA, экспрессируемых в клетках посредством аутофагии. Аутофагия может позволить упорядоченную деградацию и рециклирование клеточных компонентов. Аутофагия может включать макроаутофагию, микроаутофагию и опосредуемую шаперонами аутофагию. Пептиды могут презентироваться белком HLA, экспрессируемым в клетках посредством фагоцитоза. Фагоцитоз может быть основным механизмом, используемым для удаления патогенов и клеточного дебриса. Например, когда макрофаг поглощает патогенный микроорганизм, патоген заключается в фагосому, которая затем сливается с лизосомой с формированием фаголизосомы. В HLA класса II фагоциты, такие как макрофаги и незрелые дендритные клетки, могут захватывать объекты посредством фагоцитоза в фагосомы - хотя B-клетки демонстрируют более общий эндоцитоз в эндосомы - которые могут сливаться с лизосомами, кислые ферменты которых расщепляют захваченный белок на множество различных пептидов.
[0528] Качество обучающих данных может возрастать при использовании множества качественных показателей. Множество качественных показателей могут включать общее устранение контаминирующих пептидов, высокую оцененную интенсивность пика, высокую оценку и высокую точность массы. Перед проведением оценки можно использовать оцененную интенсивность пика. MS/MS Search сначала осуществляет скрининг спектра MS/MS против последовательностей-кандидатов с использованием простого фильтра. Этот фильтр может представлять собой минимальную оцененную интенсивность пика. Использование оцененной интенсивности пика может усиливать скорость поиска, позволяя быструю и упрощенную отбраковку последовательностей-кандидатов после изучения достаточного количества спектрального пика и обнаружения, что они не удовлетворяют пороговому значению, определяемому фильтром. Оцененная интенсивность пика может составлять по меньшей мере 50%. Оцененная интенсивность пика может составлять по меньшей мере 70%. Оцененная интенсивность пика может составлять по меньшей мере 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90% или более. В некоторых случаях оцененная интенсивность пика может составлять не более 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20%, 10% или менее. Показатель может составлять по меньшей мере 7. Оценка может составлять по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20 или более. В некоторых случаях показатель может составлять не более чем приблизительно 20, 15, 10, 9, 8, 7, 6, 5, 4, 3, 2, 1 или менее. Точность массы может составлять не более 5 м.д. Точность массы может составлять не более 10 м.д., 9 м.д., 8 м.д., 7 м.д., 6 м.д., 5 м.д., 4 м.д., 3 м.д., 2 м.д., 1 м.д. или менее. Точность массы может составлять по меньшей мере 1 м.д., 2 м.д., 3 м.д., 4 м.д., 5 м.д., 6 м.д., 7 м.д., 8 м.д., 9 м.д., 10 м.д. или более.
[0529] В некоторых вариантах осуществления точность массы составляет не более 2 м.д. В некоторых вариантах осуществления показатель расщепления основной составляет по меньшей мере 5. В некоторых вариантах осуществления показатель расщепления основной цепи составляет по меньшей мере 8.
[0530] Пептиды, презентируемые белком HLA, экспрессируемым в клетках, могут представлять собой пептиды, презентируемые единичным иммунопреципитированным белком HLA, экспрессируемым в клетках. Иммунопреципитация (IP) может представлять собой способ преципитации белкового антигена из раствора с использованием антитела, которое специфически связывается с этим конкретным белком. Этот процесс может использоваться для выделения и концентрирования конкретного белка из образца, содержащего множество тысяч различных белков. Иммунопреципитация может требовать связывания антитела с твердой подложкой в некоторый момент процедуры.
[0531] Пептиды, презентируемые белком HLA, экспрессируемым в клетках, могут представлять собой пептиды, презентируемые единичным экзогенным белком HLA, экспрессируемым в клетках. Единичный экзогенный белок HLA может быть получен путем введения одного или более экзогенных пептидов в популяцию клеток. В некоторых вариантах осуществления введение включает приведение популяции клеток в контакт с одним или более экзогенными пептидами или экспрессию одного или более экзогенных пептидов в популяции клеток. В некоторых вариантах осуществления введение включает приведение популяции клеток в контакт с одной или более нуклеиновыми кислотами, кодирующими один или более экзогенных пептидов. В некоторых вариантах осуществления одна или более нуклеиновых кислот, кодирующих один или более пептидов, представляет собой ДНК. В некоторых вариантах осуществления одна или более нуклеиновых кислот, кодирующих один или более пептидов, представляет собой РНК, необязательно где РНК представляет собой мРНК. В некоторых вариантах осуществления обогащение не включает использование тетрамерого (или мультимерного) реагента.
[0532] Пептиды, презентируемые белком HLA, экспрессируемым в клетках, могут представлять собой пептиды, презентируемые единичным рекомбинантным белком HLA, экспрессируемым в клетках. Рекомбинантный белок HLA может кодироваться рекомбинантным аллелем HLA класса I или HLA класса II. HLA класса I может быть выбран из группы, состоящей из HLA-A, HLA-B, HLA-C. HLA класса I может представлять собой неклассическую группу класса I-b. HLA класса I может быть выбран из группы, состоящей из HLA-E, HLA-F и HLA-G. HLA класса I может представлять собой представитель неклассической группы класса I-b, выбранный из группы, состоящей из HLA-E, HLA-F и HLA-G. В некоторых вариантах осуществления HLA класса II включает α-цепь HLA класса II, β-цепь HLA класса II или их комбинацию.
[0533] Множество прогностических переменных может включать прогностическую переменную аффинности пептид-HLA. Множество прогностических переменных может включать прогностическую переменную уровня экспрессии исходного белка. Уровень экспрессии исходного белка может представлять собой уровень экспрессии исходного белка пептида в клетке. В некоторых вариантах осуществления уровень экспрессии может быть определен посредством измерения количества исходного белка или количества РНК, кодирующей белок-источник. Множество прогностических переменных может включать пептидную последовательность, физические свойства аминокислот, физические свойства пептидов, уровень экспрессии исходного белка пептида в клетке, стабильность белка, уровень трансляции белка, участки убиквитинилирования, скорость деградации белка, эффективность трансляции на основе определения рибосомального профиля, способность белка к расщеплению, локализацию белка, мотивы белка хозяина, которые облегчают транспорт TAP, белок хозяина, подвергаемый аутофагии, мотивы которые способствуют остановке рибосом (например, полипролиновые или полилизиновые участки), белковые признаки, которые способствуют NMD (например, длинный 3' UTR, стоп-кодон на >50 нт выше последней точки соединения экзон:экзон и способность пептида к расщеплению).
[0534] Множество прогностических переменных может включать прогностическую переменную способности пептида к расщеплению. Способность пептида к расщеплению может быть ассоциирована с расщепляемым линкером или последовательностью расщепления. В некоторых вариантах осуществления расщепляемый линкер представляет собой участок рибосомального пропускания или элемент внутреннего участка связывания рибосомы (IRES). В некоторых вариантах осуществления участок рибосомального пропускания или IRES расщепляется при экспрессии в клетках. В некоторых вариантах осуществления участок рибосомального пропускания выбран из группы, состоящей из F2A, T2A, P2A и E2A. В некоторых вариантах осуществления элемент IRES выбран из обычных клеточных или вирусных последовательностей IRES. Последовательность расщепления, такая как F2A или участок внутренней посадки рибосомы (IRES), может находиться между α-цепью и β2-микроглобулином (HLA класса I) или между α-цепью и β-цепью (HLA класса II). В некоторых вариантах осуществления единичный аллель HLA класса I представляет собой HLA-A*02:01, HLA-A*23:01 и HLA-B*14:02 или HLA-E*01:01, и аллель HLA класса II представляет собой HLA-DRB*01:01, HLA-DRB*01:02 и HLA-DRB*11:01, HLA-DRB*15:01 или HLA-DRB*07:01. В некоторых вариантах осуществления последовательность расщепления представляет собой последовательность T2A, P2A, E2A или F2A. Например, последовательность расщепления может представлять собой E G R G S L T C G D V E N P G P (SEQ ID NO: 6) (T2A), A T N F S L K Q A G D V E N P G P (SEQ ID NO: 7) (P2A), Q C T N Y A L K L A G D V E S N P G P (SEQ ID NO: 8) (E2A) или V K Q T L N F D L K L A G D V E S N P G P (SEQ ID NO: 9) (F2A).
[0535] В некоторых вариантах осуществления последовательность расщепления может представлять собой участок расщепления тромбином CLIP.
[0536] Пептиды, презентируемые белком HLA, могут включать пептиды, идентифицированные посредством поиска по отсутствию ферментной специфичности в базе данных пептидов без модификации. Пептидная база данных может представлять собой базу данных пептидов без ферментной специфичности, такую как база данных с модификациями или база данных без модификаций (например, фосфорилирование или цистеинилирование). В некоторых вариантах осуществления пептидная база данных представляет собой базу данных полипептидов. В некоторых вариантах осуществления база данных полипептидов может представлять собой базу данных белков. В некоторых вариантах осуществления способ дополнительно включает поиск в базе данных пептидов с использованием стратегии обратного поиска в базе данных. В некоторых вариантах осуществления способ дополнительно включает поиск в базе данных белков с использованием стратегии обратного поиска в базе данных. В некоторых вариантах осуществления проводят поиск de novo, например, для обнаружения новых пептидов, которые не включены в нормальную базу данных пептидов или белков. База данных пептидов может быть получена путем предоставления первой и второй клеточных популяций, каждая из которых содержит одну или более клеток, содержащих HLA, меченный аффинной акцепторной меткой, где последовательность HLA, меченного аффинной акцепторной меткой, содержит отличающийся рекомбинантный полипептид, кодируемый отличающимся аллелем HLA, функционально связанным с аффинным акцепторным пептидом; обогащения комплексов меченный аффинной акцепторной меткой HLA-пептид; охарактеризации пептида или его части, связанной с комплексом меченный аффинной акцепторной меткой HLA-пептид после обогащения; и получения базы данных HLA-аллелеспецифический пептид.
[0537] Пептиды, презентируемые белком HLA, могут включать пептиды, идентифицированные посредством сравнения спектров MS/MS HLA-пептидов со спектрами MS/MS одного или более HLA-пептидов в базе данных пептидов.
[0538] Как на пептидах, так и на нуклеиновой кислоте, которая кодирует пептиды, может существовать мутация. Мутация может быть выбрана из группы, состоящей из точковой мутации, мутации участка сплайсинга, мутации со сдвигом рамки считывания, мутации со сквозным прочитыванием и мутации со слиянием генов. Точковая мутация может представлять собой генетическую мутацию, где единичное нуклеотидное основание изменено, встроено или удалено из последовательности ДНК или РНК. Мутация участка сплайсинга может представлять собой генетическую мутацию, которая встраивает, удаляет или изменяет количество нуклеотидов в конкретном участке, в котором происходит сплайсинг в ходе процессинга матричной РНК-предшественника в зрелую матричную РНК. Мутация со сдвигом рамки считывания может представлять собой генетическую мутацию, вызываемую инсерциями-делециями (инсерции или делеции) количества нуклеотидов в последовательности ДНК, которое не делится на три. Мутация также может включать инсерции, делеции, мутации с заменами, дупликации генов, хромосомные транслокации и хромосомные инверсии.
[0539] В некоторых вариантах осуществления белок HLA класса II включает белок HLA-DR.
[0540] В некоторых вариантах осуществления белок HLA класса II включает белок HLA-DP.
[0541] В некоторых вариантах осуществления белок HLA класса II включает белок HLA-DQ.
[0542] В некоторых вариантах осуществления белок HLA класса II может быть выбран из группы, состоящей из белка HLA-DR, и HLA-DP или HLA-DQ. В некоторых вариантах осуществления белок HLA представляет собой белок HLA класса II, выбранный из группы, состоящей из: HLA-DPB1*01:01/HLA-DPA1*01:03, HLA-DPB1*02:01/HLA-DPA1*01:03, HLA-DPB1*03:01/HLA-DPA1*01:03, HLA-DPB1*04:01/HLA-DPA1*01:03, HLA-DPB1*04:02/HLA-DPA1*01:03, HLA-DPB1*06:01/HLA-DPA1*01:03,HLA-DQB1*02:01/HLA-DQA1*05:01,HLA-DQB1*02:02/HLA-DQA1*02:01, HLA-DQB1*06:02/HLA-DQA1*01:02,HLA-DQB1*06:04/HLA-DQA1*01:02, HLA-DRB1*01:01, HLA-DRB1*01:02, HLA-DRB1*03:01, HLA-DRB1*03:02, HLA-DRB1*04:01, HLA-DRB1*04:02, HLA-DRB1*04:03, HLA-DRB1*04:04, HLA-DRB1*04:05, HLA-DRB1*04:07, HLA-DRB1*07:01, HLA-DRB1*08:01, HLA-DRB1*08:02, HLA-DRB1*08:03, HLA-DRB1*08:04, HLA-DRB1*09:01, HLA-DRB1*10:01, HLA-DRB1*11:01, HLA-DRB1*11:02, HLA-DRB1*11:04, HLA-DRB1*12:01, HLA-DRB1*12:02, HLA-DRB1*13:01, HLA-DRB1*13:02, HLA-DRB1*13:03, HLA-DRB1*14:01, HLA-DRB1*15:01, HLA-DRB1*15:02, HLA-DRB1*15:03, HLA-DRB1*16:01, HLA-DRB3*01:01, HLA-DRB3*02:02, HLA-DRB3*03:01, HLA-DRB4*01:01, HLA-DRB5*01:01). Пептиды, презентируемые белком HLA, могут иметь длину 15-40 аминокислот. Пептиды, презентируемые белком HLA, могут иметь длину по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27 или более аминокислот. В некоторых вариантах осуществления пептиды, презентируемые белком HLA, могут иметь длину не более 30, 29, 28, 27, 26, 25, 24, 23, 22, 21, 20, 19, 18, 17, 16, 15, 14, 13, 12, 11 или менее аминокислот.
[0543] Пептиды, презентируемые белком HLA, могут включать пептиды, идентифицированные посредством (a) выделения одного или более комплексов HLA из клеточной линии, экспрессирующей единичный аллель HLA класса II; (b) выделения одного или более HLA-пептидов из одного или более выделенных комплексов HLA; (c) получения спектров MS/MS для одного или более выделенных HLA-пептидов; и (d) получения пептидной последовательности, которая соответствует спектрам MS/MS одного или более выделенных HLA-пептидов из базы данных пептидов; где одна или более последовательностей, полученных на стадиях (a, b, c) и (d), идентифицирует последовательность одного или более выделенных HLA-пептидов.
[0544] Выделение может включать выделение комплексов HLA-пептид из клеток, трансфицированных или трансдуцированных конструкциями HLA, меченными аффинными метками. В некоторых вариантах осуществления комплексы могут быть выделены с использованием стандартных способов иммунопреципитации, известных в данной области, с использованием коммерчески доступных антител. Сначала клетки можно подвергать лизису. Комплексы HLA класса II-пептид можно выделять с использованием антител, специфичных к HLA класса II, таких как моноклональное антитело M5/114.15.2. В некоторых вариантах осуществления один (или пара) аллелей HLA экспрессируются в качестве слитого белка с пептидной меткой, и комплексы HLA-пептид выделяют с использованием связывающих молекул, которые распознают пептидные метки.
[0545] Выделение может включать выделение пептидов из комплексов HLA-пептид и секвенирование пептидов. Пептиды выделяют из комплекса любым известным способом, известным специалисту в данной области, таким как кислотное элюирование. Хотя можно использовать любой способ секвенирования, в некоторых вариантах осуществления используются способы с использованием масс-спектрометрии, такие как жидкостная хроматография-масс-спектрометрия (LC-MS или LC-MS/MS, или альтернативно ВЭЖХ-MS или ВЭЖХ-MS/MS). Эти способы секвенирования могут быть хорошо известны специалисту в данной области и рассмотрены в Medzihradszky KF and Chalkley RJ. Mass Spectrom Rev. 2015 Jan-Feb; 34(1):43-63.
[0546] Дополнительные компоненты-кандидаты и молекулы, пригодные для выделения или очистки, могут включать связывающие молекулы, такие как биотин (специфическая связывающаяся пара биотин-авидин), антитело, рецептор, лиганд, лектин или молекулы, которые содержат твердую подложку, включая, например, пластмассовые или полистироловые гранулы, чашки или гранулы, магнитные гранулы, тест-полоски и мембраны. Для разделения конъюгатов по различию зарядов можно использовать способы очистки, такие как катионообменная хроматография, которые эффективно разделяют конъюгаты по различиям в молекулярной массе. Содержимое фракций, полученных посредством катионообменной хроматографии, можно идентифицировать по молекулярной массе с использованием общепринятых способов, например, масс-спектроскопии, SDS-PAGE или других известных способов разделения молекулярных структур по молекулярной массе.
[0547] В некоторых вариантах осуществления способ дополнительно включает выделение пептидов и меченных аффинной акцепторной меткой комплексов HLA-пептид перед охарактеризацией. В некоторых вариантах осуществления комплекс HLA-пептид выделяют с использованием антитела против HLA. В некоторых случаях комплекс HLA-пептид с аффинной меткой или без нее выделяют с использованием антитела против HLA. В некоторых случаях растворимый HLA (sHLA) с аффинной меткой или без нее выделяют из среды клеточной культуры. В некоторых случаях растворимый HLA (sHLA) с аффинной меткой или без нее выделяют с использованием антитела против HLA. Например, HLA, такой как растворимый HLA (sHLA), с аффинной меткой или без нее, можно выделять с использованием гранулы или колонки, содержащей антитело против HLA. В некоторых вариантах осуществления пептиды выделяют с использованием антител против HLA. В некоторых случаях растворимый HLA (sHLA) с аффинной меткой выделяют с использованием антитела против HLA. В некоторых случаях растворимый HLA (sHLA) с аффинной меткой или без нее выделяют с использованием колонки, содержащей антитело против HLA. В некоторых вариантах осуществления способ дополнительно включает удаление одной или более аминокислот с конца пептида, связанного с меченным аффинной акцепторной меткой комплексом HLA-пептид.
[0548] Кроме того, персонализированная вакцина против злокачественной опухоли может содержать адъювант. Например, поли-ICLC, агонист TLR3 и доменов РНК хеликазы MDA5 и RIG3, продемонстрировал несколько желаемых свойств для адъюванта вакцины. Эти свойства могут включать индукцию локальной и системной активации иммунных клеток in vivo, продуцирование стимулирующих хемокинов и цитокинов, и стимуляцию презентации антигенов посредством DC. Более того, поли-ICLC может индуцировать длительные ответы CD4+ и CD8+ у человека. Важно, что поразительные сходства в активации транскрипции и передачи сигнала могут наблюдаться у индивидуумов, вакцинированных посредством поли-ICLC, и у добровольцев, которым вводили высокоэффективную репликационно-компетентную вакцину против желтой лихорадки. Более того, >90% пациентов с карциномой яичника, иммунизированных поли-ICLC в комбинации с пептидной вакциной NYESO-1 (в дополнение к Montanide) продемонстрировали индукцию CD4+ и CD8+ T-клеток, а также антительные ответы на пептид в недавнем испытании фазы 1.
[0549] Кроме того, персонализированная вакцина против злокачественной опухоли, может содержать ингибитор иммунной точки контроля. Ингибитор иммунной точки контроля может включать тип лекарственного средства, которое блокирует определенные белки, продуцированные некоторыми типами клеток иммунной системы, такими как T-клетки и некоторые злокачественные клетки. Эти белки помогают держать иммунные ответы под контролем и могут удерживать T-клетки от уничтожения злокачественных клеток. Когда эти белки блокируются, "тормоза" иммунной системы отпускаются и T-клетки способны лучше уничтожать злокачественные клетки. Примеры белков точки контроля, присутствующих на T-клетках или злокачественных клетках, включают PD-1/PD-L1 и CTLA-4/B7-1/B7-2. Некоторые ингибиторы иммунной точки контроля используются для лечения злокачественной опухоли.
[0550] Обучающие данные, кроме того, могут включать структурированные данные, данные временного ряда, неструктурированные данные и реляционные данные. Неструктурированные данные могут включать аудиоданные, данные в виде изображений, видео, механические данные, электрические данные, химические данные и любую их комбинацию, для применения в точной имитации или обучении робототехники или имитационных моделей. Данные временного ряда могут включать данные от одного или более из интеллектуального счетчика, интеллектуального приспособления, интеллектуального устройства, системы мониторинга, устройства телеметрии или датчика. Реляционные данные включают данные системы потребителя, системы масштаба предприятия, операционной системы, веб-сайта, доступного через сеть прикладного программного интерфейса (API) или любой их комбинации. Введение данных может осуществлять пользователь посредством любого способа ввода файлов или других форматов данных в программное обеспечение или системы.
[0551] Обучающие данные могут быть загружены в базу данных на основе облака. К базе данных на основе облака может быть осуществлен доступ из локальных и/или удаленных компьютерных систем, на которых запускаются алгоритмы обработки сигнала датчика на основе обучения. База данных на основе облака и ассоциированное с ней программное обеспечение могут использоваться для архивации электронных данных, распространения электронных данных и анализа электронных данных. Данные или наборы данных, полученные локально, могут быть загружены в базу данных на основе облака, из которой к ним может быть получен доступ, и они могут быть использованы для обучения других машинных систем детекции на основе обучения в том же месте или другом месте. Результаты тестов сенсорного устройства и системы, полученные локально, могут быть загружены в базу данных на основе облака и использованы для обновления обучающего набора данных в реальном времени для непрерывного усовершенствования эффективности тестирования сенсорного устройства и системы детекции.
[0552] Обучение можно проводить с использованием сверточных нейронных сетей. Сверточная нейронная сеть (CNN) описана в настоящем описании. Сверточные нейронные сети могут включать по меньшей мере два сверточных слоя. Количество сверточных слоев может составлять 1-10, и количество расширенных слоев может составлять 0-10. Общее количество сверточных слоев (включая входной и выходной слои) может составлять по меньшей мере приблизительно 1,2, 3, 4, 5, 10, 15, 20 или более, и общее количество расширенных слоев может составлять по меньшей мере приблизительно 1,2, 3, 4, 5, 10, 15, 20 или более. Общее количество сверточных слоев может составлять не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее, и общее количество расширенных слоев может составлять не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее. В некоторых вариантах осуществления количество сверточных слоев составляет 1-10, и количество полносвязных слоев составляет 0-10. Общее количество сверточных слоев (включая входной и выходной слои) может составлять по меньшей мере приблизительно 1,2, 3, 4, 5, 10, 15, 20 или более, и общее количество полносвязных слоев может составлять по меньшей мере приблизительно 1,2, 3, 4, 5, 10, 15, 20 или более. Общее количество сверточных слоев может составлять не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее, и общее количество полносвязных слоев может составлять не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее.
[0553] Сверточные нейронные сети может включать по меньшей мере одну стадию пакетной нормализации. Слой пакетной нормализации может повышать эффективность и стабильность нейронных сетей. Слой пакетной нормализации может представлять собой любой слой нейронной сети с входными данными, которые имеют нулевую среднюю/единичную дисперсию. Общее количество слоев пакетной нормализации может составлять по меньшей мере приблизительно 3, 4, 5, 10, 15, 20 или более. Общее количество слоев пакетной нормализации может составлять не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее.
[0554] Сверточные нейронные сети могут включать по меньшей мере одну стадию пространственного исключения. Общее количество стадий пространственного исключения может составлять по меньшей мере приблизительно 3, 4, 5, 10, 15, 20 или более, и общее количество стадий пространственного исключения может составлять не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее.
[0555] Сверточные нейронные сети могут включать по меньшей мере одну стадию глобального максимального пулинга. Слои глобального пулинга могут объединять результаты нейронных кластеров одного слоя в один нейрон следующего слоя. Например, слои максимального пулинга могут использовать максимальную величину из каждого кластера нейронов на предыдущем слое; и слои усредненного пулинга могут использовать среднюю величину из каждого кластера нейронов на предыдущем слое. Сверточные нейронные сети могут включать по меньшей мере приблизительно 1, 2, 3, 4, 5, 10, 15, 20 или более стадий глобального максимального пулинга. Сверточные нейронные сети могут включать не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее стадий глобального максимального пулинга.
[0556] Сверточные нейронные сети могут включать по меньшей мере один плотный слой. Сверточные нейронные сети могут содержать по меньшей мере приблизительно 1, 2, 3, 4, 5, 10, 15, 20 или более плотных слоев. Сверточные нейронные сети могут содержать не более чем приблизительно 20, 15, 10, 5, 4, 3 или менее плотных слоев.
Способы терапии
[0557] Персонализированная иммунотерапия с использованием опухолеспецифических пептидов была описана. Опухолевые неоантигены, которые появляются в результате генетического изменения (например, инверсии, транслокации, делеции, миссенс-мутации, мутации участков сплайсинга и т.д.) в злокачественных клетках представляют собой наиболее опухолеспецифический класс антигенов. Неоантигены редко используются в вакцине против злокачественной опухоли или иммуногенных композициях вследствие технически затрудненной их идентификации, выбора оптимизированных антигенов и получения неоантигенов для применения в вакцинной или иммуногенной композиции. Эффективный выбор того, какие конкретно антигены использовать в качестве иммуногена, требует возможности прогнозировать, какие опухолеспецифические пептиды будут эффективно связываться с аллелями HLA, присутствующими у пациента, и будут эффективно презентироваться иммунной системе пациента для индукции противоопухолевого иммунитета. Одним из ключевых препятствий для разработки излечивающей и опухолеспецифической иммунотерапии является идентификация и селекция высокоспецифических и рестрицированных опухолевых антигенов, чтобы избежать аутоиммунитета. Это является особенно важным в случае опухолеспецифических пептидов, являющихся кандидатами для иммунотерапии, которые презентируются антигенами MHC класса II, поскольку существует определенный уровень неизбирательности в отношении связывания MHC класса II-пептид и презентации его иммунной системе. В то же время презентируемые MHC класса II пептиды требуются для активации не только цитотоксических клеток, но также CD4+ T-клеток памяти. Таким образом, опосредуемый MHC класса II иммунногенный ответ необходим для устойчивого ответа, обеспечивая долговременную иммуногенность для большей эффективности защиты против опухоли. Эти проблемы могут решаться посредством: наличия надежного алгоритма прогнозирования пептид-MHC и наличие надежной системы для анализа и валидации взаимодействия пептид-MHC и иммуногенности. Таким образом, в некоторых вариантах осуществления высокоэффективная и иммуногенная вакцина против злокачественной опухоли может быть получена путем идентификации мутаций-кандидатов в новообразованиях/опухолях, которые присутствуют в опухоли на уровне ДНК, но не в соответствующих образцах зародышевого типа, у высокой доли индивидуумов, имеющих злокачественную опухоль; анализа идентифицированных мутаций посредством одного или более алгоритмов прогнозирования связывания пептид-MHC для идентификации того, какой MHC (лейкоцитарный антиген человека или HLA в случае людей) связывается с высокой долей аллелей HLA пациента; и синтеза множества неоантигенный пептидов, выбранных из наборов всех неоантигенных пептидов и спрогнозированных связывающих пептидов, для применения в вакцине против злокачественной опухоли или иммуногенной композиции, пригодной для лечения высокой доли индивидуумов, имеющих злокачественную опухоль.
[0558] Например, транслирование информации о секвенировании пептидов в терапевтическую вакцину может включать прогнозирование мутантных пептидов, которые могут связываться с пептидами HLA у высокой доли индивидуумов. Эффективный выбор конкретных мутаций для использования в качестве иммуногена требует возможности прогнозирования того, какие мутантные пептиды будут эффективно связываться с высокой долей аллелей HLA пациентов. Недавно подходы обучения на основе нейронных сетей с валидированным связывающимися и не связывающимися пептидами увеличили точность алгоритмов прогнозирования для большинства аллелей HLA-A и -B. Однако, хотя использование продвинутых алгоритмов на основе нейронных сетей помогло установить правила связывания HLA-пептид, некоторые факторы ограничивают мощность прогнозирования пептидов, презентируемых аллелями HLA.
[0559] Например, транслирование информации о секвенировании пептидов в терапевтическую вакцину может включать составление лекарственного средства в качестве мультиэпитопной вакцины из длинных пептидов. Нацеливание на настолько много мутантных эпитопов, насколько это практически возможно, использует преимущество огромной способности иммунной системы, препятствует возможности иммунологического ускользания посредством подавления продукта иммунного гена-мишени, и компенсирует известную неточность подходов прогнозирования эпитопов. Синтетические пептиды обеспечивают полезное средство для эффективного получения множества иммуногенов и быстрой трансляции идентификации мутантных эпитопов в эффективную вакцину. Пептиды можно без труда синтезировать химически и легко очищать с использованием реагентов, свободных от контаминирующих бактерий или животных веществ. Малый размер позволяет отчетливое фокусирование на мутантной области белка, и также снижает постороннюю антигенную конкуренцию со стороны других компонентов (немутантный белок или вирусные векторные антигены).
[0560] Например, транслирование информации о секвенировании пептидов в терапевтическую вакцину может включать комбинацию с сильным вакцинным адъювантом. Эффективные вакцины могут требовать сильного адъюванта для инициации иммунного ответа. Например, поли-ICLC, агонист TLR3 и доменов РНК хеликазы MDA5 и RIG3, продемонстрировал несколько желаемых свойств для адъюванта вакцины. Эти свойства могут включать индукцию локальной и системной активации иммунных клеток in vivo, продуцирование стимулирующих хемокинов и цитокинов, и стимуляцию презентации антигенов посредством DC. Более того, поли-ICLC может индуцировать длительные ответы CD4+ и CD8+ у человека. Важно, что поразительные сходства в активации транскрипции и передачи сигнала могут наблюдаться у индивидуумов, вакцинированных посредством поли-ICLC, и у добровольцев, которым вводили высокоэффективную репликационно-компетентную вакцину против желтой лихорадки. Более того, >90% пациентов с карциномой яичника, иммунизированных поли-ICLC в комбинации с пептидной вакциной NYESO-1 (в дополнение к Montanide), продемонстрировали индукцию CD4+ и CD8+ T-клеток, а также антительные ответы на пептид в недавнем испытании фазы 1. В то же время на сегодняшний день поли-ICLC был тщательно протестирован в более чем 25 клинических испытаниях и продемонстрировал относительно благоприятный профиль токсичности.
[0561] В некоторых вариантах осуществления иммуногенные пептиды можно идентифицировать из клеток индивидуума с заболеванием или состоянием. В некоторых вариантах осуществления иммуногенные пептиды могут быть специфическими для индивидуума с заболеванием или состоянием. В некоторых вариантах осуществления иммуногенные пептиды могут связываться с HLA, который соответствует гаплотипу HLA индивидуума с заболеванием или состоянием.
[0562] В некоторых вариантах осуществления библиотеку пептидов можно экспрессировать в клетках. В некоторых вариантах осуществления клетки включают пептиды, подлежащие идентификации или охарактеризации. В некоторых вариантах осуществления пептиды, подлежащие идентификации или охарактеризации, представляют собой эндогенные пептиды. В некоторых вариантах осуществления пептиды представляют собой экзогенные пептиды. Например, пептиды, подлежащие идентификации или охарактеризации, могут экспрессироваться с множества последовательностей, кодирующих библиотеку пептидов.
[0563] До предоставления настоящего описания в большинстве исследований посредством LC-MS/MS пептидома HLA использовались клетки, экспрессирующие множество пептидов HLA, что требовало отнесения пептидов к 1 из вплоть до 6 аллелей HLA класса I с использованием ранее существовавших биоинформатических средств прогнозирования или "обратной свертки" (Bassani-Sternberg and Gfeller, 2016). Таким образом, пептиды, которые не имеют высокого соответствия известным мотивам, не могли быть уверенно описаны в качестве связывающихся с данным аллелем HLA.
[0564] В рамках настоящего изобретения предусматриваются способы прогнозирования пептидов, таких как мутантные пептиды, которые связываются с пептидами HLA индивидуумов. В некоторых вариантах осуществления заявка относится к способам идентификации из данного набора содержащих антиген пептидов наиболее подходящих пептидов для получения иммуногенной композиции для индивидуума, причем указанный способ включает выбор из данного набора пептидов множества пептидов, способных связывать белок HLA индивидуума, где указанная способность связывать белок HLA определяется путем анализа последовательности пептидов с использованием устройства, обученного с использованием баз данных последовательностей пептидов, соответствующих конкретным HLA-связывающим пептидам для каждого из аллелей HLA указанного индивидуума. В рамках настоящего изобретения предусматриваются способы идентификации из данного набора содержащих антиген пептидов наиболее подходящих пептидов для получения иммуногенной композиции для индивидуума, причем указанный способ включает выбор из данного набора пептидов множества пептидов, определенных в качестве способных связывать белок HLA индивидуума, причем способность связывать белок HLA определяют посредством анализа последовательности пептидов с использованием устройства, которое обучено с использованием базы данных последовательностей пептидов, полученной путем выполнения способов, описанных в настоящем описании выше. Таким образом, в некоторых вариантах осуществления настоящее изобретение относится к способам идентификации множества специфических для индивидуума пептидов для получения специфической для индивидуума иммуногенной композиции, где индивидуум имеет опухоль, и специфические для опухоли пептиды являются специфическими для индивидуума и опухоли индивидуума, причем указанный способ включает: секвенирование образца опухоли индивидуума и неопухолевого образца индивидуума; определение на основе секвенирования нуклеиновых кислот: немолчащих мутаций, присутствующих в геноме злокачественных клеток индивидуума, но не в нормальной ткани индивидуума, и HLA-генотипа индивидуума; и выбор на основе идентифицированных немолчащих мутаций множества специфических для индивидуума пептидов, причем каждый из них имеет отличающийся опухолевый эпитоп, специфический для опухоли индивидуума, и каждый из них идентифицирован как способный связывать белок HLA индивидуума при определении посредством анализа последовательности пептидов, образовавшихся в результате немолчащих мутаций, в способах прогнозирования связывания HLA, описанных в настоящем описании.
[0565] В некоторых вариантах осуществления в настоящем описании описан способ охарактеризации комплексов HLA-пептид, специфических для индивидуума.
[0566] В некоторых вариантах осуществления способ охарактеризации комплексов HLA-пептид, специфических для индивидуума, используют для разработки иммунотерапевтического средства для индивидуума, нуждающегося в этом, такого как индивидуум с состоянием или заболеванием.
[0567] В рамках настоящего изобретения предусматривается способ обеспечения противоопухолевого иммунитета у млекопитающего, включающий введение млекопитающему полинуклеиновой кислоты, содержащей последовательность, кодирующую пептид, идентифицированный в соответствии с описанным способом. В рамках настоящего изобретения предусматривается способ обеспечения противоопухолевого иммунитета у млекопитающего, включающий введение млекопитающему эффективного количества пептида с последовательностью пептида, идентифицированной способом, описанным в настоящем описании. В рамках настоящего изобретения предусматривается способ обеспечения противоопухолевого иммунитета у млекопитающего, включающий введение млекопитающему клетки, содержащей пептид, содержащий последовательность пептида, идентифицированную способом, описанным в настоящем описании. В рамках настоящего изобретения предусматривается способ обеспечения противоопухолевого иммунитета у млекопитающего, включающий введение млекопитающему клетки, содержащей полинуклеиновую кислоту, содержащую последовательность, кодирующую пептид, содержащий последовательность пептида, идентифицированного способом, описанным в настоящем описании. В некоторых вариантах осуществления клетка презентирует пептид в качестве комплекса HLA-пептид.
[0568] В рамках настоящего изобретения предусматривается способ лечения заболевания или нарушения у индивидуума, причем способ включает введение индивидууму полинуклеиновой кислоты, содержащей последовательность, кодирующую пептид, идентифицированный способом, описанным в настоящем описании. В рамках настоящего изобретения предусматривается способ лечения заболевания или нарушения у индивидуума, причем способ включает введение индивидууму эффективного количества пептида, содержащего последовательность пептида, идентифицированного способом, описанным в настоящем описании. В рамках настоящего изобретения предусматривается способ лечения заболевания или нарушения у индивидуума, причем способ включает введение индивидууму клетки, содержащий пептид, содержащий последовательность пептида, идентифицированного способом, описанным в настоящем описании. В рамках настоящего изобретения предусматривается способ лечения заболевания или нарушения у индивидуума, причем способ включает введение индивидууму клетки, содержащей полинуклеиновую кислоту, содержащую последовательность, кодирующую пептид, содержащий последовательность пептида, идентифицированного способом, описанным в настоящем описании. В некоторых вариантах осуществления заболевание или нарушение представляет собой злокачественную опухоль. В некоторых вариантах осуществления способ дополнительно включает введение индивидууму ингибитора иммунной точки контроля.
[0569] В некоторых вариантах осуществления в рамках настоящего изобретения описаны способы разработки иммунотерапевтического средства для индивидуума, нуждающегося в этом, посредством охарактеризации комплексов HLA-пептид, включающие: a) предоставление популяции клеток, полученных от индивидуума, нуждающегося в этом, где одна или более клеток в популяции клеток содержат полинуклеиновую кислоту, содержащую последовательность, кодирующую меченный аффинной акцепторной меткой аллель HLA класса I или HLA класса II, где последовательность, кодирующая меченный аффинной акцепторной меткой HLA, содержит: i) последовательность, кодирующую рекомбинантный аллель HLA класса I или HLA класса II, функционально связанный с ii) последовательностью, кодирующей аффинный акцепторный пептид; b) экспрессию меченного аффинной акцепторной меткой HLA по меньшей мере в одной клетке из одной или более клеток популяции клеток, тем самым формируя комплексы меченный аффинной акцепторной клеткой HLA-пептид по меньшей мере в одной клетке; c) увеличение содержания комплексов меченный аффинной акцепторной меткой HLA-пептид, охарактеризацию комплексов HLA-пептид, специфических для индивидуума, нуждающегося в этом; и d) разработку иммунотерапевтического средства на основе комплекса HLA-пептид, специфического для индивидуума, нуждающегося в этом; где индивидуум имеет заболевание или состояние.
[0570] В некоторых вариантах осуществления иммунотерапевтическое средство представляет собой терапевтическое средство на основе нуклеиновой кислоты или пептида.
[0571] В некоторых вариантах осуществления способ включает введение одного или более пептидов в популяцию клеток. В некоторых вариантах осуществления способ включает приведение популяции клеток в контакт с одним или более пептидами, экспрессирующими один или более пептидов в популяции клеток. В некоторых вариантах осуществления способ включает приведение популяции клеток в контакт с одной или более нуклеиновыми кислотами, кодирующими один или более пептидов.
[0572] В некоторых вариантах осуществления способ включает разработку иммунотерапевтического средства на основе пептидов, идентифицированных применительно к пациент-специфическим HLA. В некоторых вариантах осуществления популяция клеток получена от индивидуума, нуждающегося в этом.
[0573] В некоторых вариантах осуществления способ включает экспрессию библиотеки пептидов в популяции клеток. В некоторых вариантах осуществления способ включает экспрессию библиотеки меченных аффинной акцепторной меткой комплексов HLA-пептид. В некоторых вариантах осуществления библиотека включает библиотеку пептидов, ассоциированных с заболеванием или состоянием. В некоторых вариантах осуществления заболевание или состояние представляет собой злокачественную опухоль или инфекцию инфекционным агентом, или аутоиммунное заболевание. В некоторых вариантах осуществления способ включает введение инфекционного агента или его частей в одну или более клеток в популяции клеток. В некоторых вариантах осуществления способ включает охарактеризацию одного или более пептидов из комплекса HLA-пептид, специфических для индивидуума, нуждающегося в этом, необязательно, где пептиды происходят из одного или более белков-мишеней инфекционного агента или аутоиммунного заболевания. В некоторых вариантах осуществления способ включает охарактеризацию одной или более областей пептидов из одного или более белков-мишеней инфекционного агента или аутоиммунного заболевания. В некоторых вариантах осуществления способ включает идентификацию пептидов из комплексов HLA-пептид, образованных в результате инфекционного агента или аутоиммунного заболевания.
[0574] В некоторых вариантах осуществления инфекционный агент представляет собой патоген. В некоторых вариантах осуществления патоген представляет собой вирус, бактерию или паразит.
[0575] В некоторых вариантах осуществления вирус выбран из группы, состоящей из: вируса BK (BKV), вирусов Денге (DENV-1, DENV-2, DENV-3, DENV-4, DENV-5), цитомегаловируса (CMV), вирус гепатита B (HBV), вируса гепатита C (HCV), вируса Эпштейна-Барр (EBV), аденовируса, вируса иммунодефицита человека (ВИЧ), T-клеточного лимфотропного вируса человека (HTLV-1), вируса гриппа, RSV, HPV, вируса бешенства, вируса свинки, вируса краснухи, вируса полиомиелита, вируса желтой лихорадки, вируса гепатита A, гепатита B, ротавируса, вируса ветрянки, вируса папилломы человека (HPV), вируса натуральной оспы, вируса опоясывающего лишая и их комбинаций.
[0576] В некоторых вариантах осуществления бактерии выбраны из группы, состоящей из: Klebsiella spp., Tropheryma whipplei, Mycobacterium leprae, Mycobacterium lepromatosis и Mycobacterium tuberculosis. В некоторых вариантах осуществления бактерии выбраны из группы, состоящей из: возбудителя брюшного тифа, пневмококка, менингококка, гемоглобинофильных бактерий B, возбудителя сибирской язвы, столбнячного токсоида, менингококков группы B, БЦЖ, возбудителя холеры и их комбинаций.
[0577] В некоторых вариантах осуществления паразитом является гельминт или простейшее. В некоторых вариантах осуществления паразит выбран из группы, состоящей из: Leishmania spp. (например, L. major, L. infantum, L. braziliensis, L. donovani, L. chagasi, L. mexicana), Plasmodium spp. (например, P. falciparum, P. vivax, P. ovale, P. malariae), Trypanosoma cruzi, Ascaris lumbricoides, Trichuris trichiura, Necator americanus и Schistosoma spp. (S. mansoni, S. haematobium, S. japonicum).
[0578] В некоторых вариантах осуществления иммунотерапевтическое средство представляет собой сконструированный рецептор. В некоторых вариантах осуществления сконструированный рецептор представляет собой химерный рецептор антигена (CAR), T-клеточный рецептор (TCR) или B-клеточный рецептор (BCR), адоптивную T-клеточную терапию (ACT) или их производное. В других аспектах сконстрированный рецептор представляет собой химерный рецептор антигена (CAR). В некоторых аспектах CAR представляет собой CAR первого поколения. В других аспектах CAR представляет собой CAR второго поколения. В других аспектах CAR представляет собой CAR третьего поколения.
[0579] В некоторых аспектах CAR содержит внеклеточную часть, трансмембранную часть и внутриклеточную часть. В некоторых аспектах внутриклеточная часть содержит по меньшей мере один костимулирующий домен T-клеток. В некоторых аспектах костимулирующий домен T-клеток выбран из группы, состоящей из CD27, CD28, TNFRS9 (4-1BB), TNFRSF4 (OX40), TNFRSF8 (CD30), CD40LG (CD40L), ICOS, ITGB2 (LFA-1), CD2, CD7, KLRC2 (NKG2C), TNFRS18 (GITR), TNFRSF14 (HVEM) или любой их комбинации.
[0580] В некоторых аспектах сконструированный рецептор связывает мишень. В некоторых аспектах связывание является специфическим в отношении пептида, идентифицированного в способе охарактеризации комплексов HLA-пептид, специфических для индивидуума, страдающего от заболевания или состояния.
[0581] В некоторых аспектах иммунотерапевтическое средство представляет собой клетку, как описано в настоящем описании. В некоторых аспектах иммунотерапевтическое средство представляет собой клетку, содержащую рецептор, который специфически связывает пептид, идентифицированный посредством способа охарктеризации комплексов HLA-пептид, специфических для индивидуума, страдающего от заболевания или состояния. В некоторых аспектах иммунотерапевтическое средство представляет собой клетку, используемую в комбинации с пептидами/нуклеиновыми кислотами по настоящему изобретению. В некоторых вариантах осуществления клетка представляет собой клетку пациента. В некоторых вариантах осуществления клетка представляет собой T-клетку. В некоторых вариантах осуществления клетка представляет собой инфильтрирующий опухоль лимфоцит.
[0582] В некоторых аспектах, индивидуума с состоянием или заболеванием лечат, исходя из репертуара T-клеточных рецепторов индивидуума. В некоторых вариантах осуществления антигенную вакцину выбирают, исходя из репертуара T-клеточных рецепторов индивидуума. В некоторых вариантах осуществления индивидуума лечат T-клетками, экспрессирующими TCR, специфическими для антигена или пептида, идентифицированного с использованием способов, описанных в настоящем описании. В некоторых вариантах осуществления индивидуума лечат антигеном или пептидом, идентифицированным с использованием способов, описанных в настоящем описании, специфичным к TCR, например, специфичным к TCR индивидуума. В некоторых вариантах осуществления индивидуума лечат антигеном или пептидом, идентифицированным с использованием способов, описанных в настоящем описании, специфичным к T-клеткам, экспрессирующим TCR, например, специфичным к TCR индивидуума. В некоторых вариантах осуществления индивидуума лечат антигеном или пептидом, идентифицированным с использованием способов, описанных в настоящем описании, специфичным к TCR, специфическим для индивидуума.
[0583] В некоторых вариантах осуществления иммунногенную антигенную композицию или вакцину выбирают на основе TCR, идентифицированных у индивидуума. В одном варианте осуществления идентификацию репертуара T-клеток и тестирование его в функциональных способах анализа используют для определения иммуногенной композиции или вакцины, вводимой индивидууму с состоянием или заболеванием. В некоторых вариантах осуществления иммуногенная композиция представляет собой антигенную вакцину. В некоторых вариантах осуществления антигенная вакцина содержит специфические для индивидуума антигенные пептиды. В некоторых вариантах осуществления антигенные пептиды, подлежащие включению в антигенную вакцину, выбирают на основе количественного определения специфических для индивидуума TCR, которые связываются с антигенами. В некоторых вариантах осуществления антигенные пептиды выбирают на основе аффинности связывания пептида с TCR. В некоторых вариантах осуществления выбор основан на комбинации как количества, так и аффинности связывании. Например, TCR, который сильно связывается с антигеном в функциональном анализе, но не презентируется на высоком уровне в репертуаре TCR, может быть хорошим кандидатом для антигенной вакцины, поскольку преимущественно будут амплифицироваться T-клетки, экспрессирующие TCR.
[0584] В некоторых вариантах осуществления антигены выбирают для введения индивидууму, исходя из связывания с TCR. В некоторых вариантах осуществления T-клетки, такие как T-клетки от индивидуума с заболеванием или состоянием, могут быть увеличены в количестве. Увеличенные в количестве T-клетки, которые экспрессируют TCR, специфические к иммуногенному антигенному пептиду, идентифицированному с использованием способа, описанного в настоящем описании, можно вводить обратно индивидууму. В некоторых вариантах осуществления подходящие клетки, например PBMC, трансдуцируют или трансфицируют полинуклеотидами для экспрессии TCR, специфичных к иммуногенному антигенному пептиду, идентифицированному с использованием способа, описанному в настоящем описании, и вводят обратно индивидууму. T-клетки, экспрессирующие TCR, специфичные к иммуногенному антигенному пептиду, идентифицированному с использованием способа, описанного в настоящем описании, можно увеличивать в количестве и вводить обратно индивидууму. В некоторых вариантах осуществления T-клетки, которые экспрессируют TCR, специфичные к иммуногенному антигенному пептиду, идентифицированному с использованием способа, описанного в настоящем описании, который обеспечивает цитолитическую активность при инкубации с аутологичной пораженной заболеванием тканью, можно увеличивать в количестве и вводить индивидууму. В некоторых вариантах осуществления T-клетки, используемые в функциональном анализе, обеспечивающем связывание с иммуногенным антигенным пептидом, идентифицированным с использованием способа, описанного в настоящем описании, можно увеличивать в количестве и вводить индивидууму. В некоторых вариантах осуществления TCR, которые были определены в качестве связывающихся со специфическими для индивидуума иммуногенными антигенными пептидами, идентифицированными с использованием способа, описанного в настоящем описании, могут быть экспрессированы в T-клетках и введены индивидууму.
[0585] Способы, описанные в настоящем описании, могут вовлекать адоптивный перенос клеток иммунной системы, таких как T-клетки, специфичные к выбранным антигенам антигены, таким как ассоциированные с опухолью или патогеном антигены. Можно использовать различные стратегии для генной модификации T-клеток посредством изменения специфичности T-клеточного рецептора (TCR), например, посредством внесения новых цепей TCR α- и β со специфичностью к иммуногенному антигенному пептиду, идентифицированному с использованием способа, описанного в настоящем описании (см., например, патент США №8697854; публикации патентов PCT: WO2003020763, WO2004033685, WO2004044004, WO2005114215, WO2006000830, WO2008038002, WO2008039818, WO2004074322, WO2005113595, WO2006125962, WO2013166321, WO2013039889, WO2014018863, WO2014083173; патент США №8088379).
[0586] Химерные рецепторы антигенов (CAR) можно использовать для получения иммунореактивных клеток, таких как T-клетки, специфичные к выбранным мишеням, таким как иммуногенные антигенные пептиды, идентифицированные с использованием способа, описанного в настоящем описании, с широким множеством химерных конструкций рецептора (см., например, патенты США №5843728; 5851828; 5912170; 6004811; 6284240; 6392013; 6410014; 6753162; 8211422; и публикацию PCT W09215322). Альтернативные конструкции CAR могут быть охарактеризованы как принадлежащие к последующим поколениям. CAR первого поколения, главным образом, состоят из одноцепочечного вариабельного фрагмента антитела, специфичного к антигену, например, содержащего VL, связанную с VH специфического антитела, связанной посредством гибкого линкера, например, посредством шарнирного домена CD8a и трансмембранного домена CD8a, с трансмембранным и внутриклеточным сигнальными доменами CD3ζ, или FcRy, или scFv-FcRy (см., например, патент США №7741465; патент США №5912172; патент США №5906936). CAR второго поколения включают внутриклеточные домены одной или более костимулирующих молекул, таких как CD28, OX40 (CD134) или 4-1BB (CD137) в эндодомене, например, scFv-CD28/OX40/4-lBB-CD3 (см., например, патенты США №8911993; 8916381; 8975071; 9101584; 9102760; 9102761). CAR третьего поколения включают комбинацию костимулирующих эндодоменов, таких как сигнальные домены CD3C-цепи, CD97, GDI la-CD18, CD2, ICOS, CD27, CD154, CDS, OX40, 4-1BB или CD28, например, scFv-CD28-4-lBB-CD3C или scFv-CD28-OX40-CD3Q (см., например, патент США №8906682; патент США №8399645; патент США №5686281; публикацию PCT № WO2014134165; публикацию PCT № WO2012079000). В некоторых вариантах осуществления костимуляция может координироваться посредством экспрессии CAR в антигенспецифических T-клетках, выбранных так, чтобы они активировались или увеличивались в количестве, например, после взаимодействия с антигеном на профессиональных антигенпредставляющих клетках, при костимуляции. Дополнительные сконструированные рецепторы могут быть предоставлены на иммунорецепторных клетках, например, для усиления нацеливания T-клеточной атаки и/или минимизации побочных эффектов.
[0587] Для трансформации иммунореактивных клеток, являющихся мишенями, можно использовать альтернативные способы, такие как слияние протопластов, липофекция, трансфекция или электропорация. Для введения CAR можно использовать широкое множество векторов, таких как ретровирусные векторы, лентивирусные векторы, аденовирусные векторы, аденоассоциированные вирусные векторы, плазмиды или транспозоны, такие как траспозон Sleeping Beauty (см. патенты США №6489458; 7148203; 7160682; 7985739; 8227432), например, с использованием антигенспецифических CAR 2-го поколения, передающих сигнал через CD3ζ и либо CD28, либо CD137. Вирусные векторы могут включать, например, векторы на основе ВИЧ, SV40, EBV, HSV или BPV.
[0588] Клетки, предназначенные для трансформации, могут включать, например, T-клетки, натуральные киллеры (NK), цитотоксические T-лимфоциты (CTL), регуляторные T-клетки, эмбриональные стволовые клетки человека, инфильтрирующие опухоль лимфоциты (TIL) или плюрипотентные стволовые клетки, из которых могут дифференцироваться лимфоидные клетки. T-клетки, экспрессирующие желаемый CAR, могут быть отобраны, например, посредством сокультивирования с γ-облученными активирующими и увеличивающимися в количестве клетками (APC), которые коэкспрессируют антиген злокачественной опухоли и костимулирующие молекулы. Модифицированные CAR T-клетки могут быть увеличены в количестве, например, посредством сокультивирования на APC в присутствии растворимых факторов, таких как IL-2 и IL-21. Это увеличение в количестве можно проводить, например, чтобы получить CAR T-клетки памяти (которые, например, можно анализировать посредством неферментативной цифровой матрицы и/или мультипанельной проточной цитометрии). Таким образом, могут быть предоставлены CAR T-клетки, которые обладают специфической цитотоксической активностью против имеющих антигены опухолей (необязательно совместно с продуцированием желаемых хемокинов, таких как интерферон-γ). CAR T-клетки этого типа могут использоваться, например, в моделях на животных, например, лечения ксенотрансплантатов опухолей.
[0589] Подходы, такие как описаны выше, можно адаптировать для обеспечения способов лечения и/или увеличения выживаемости индивидуума, имеющего заболевание, такое как новообразование или патогенная инфекция, например посредством введения эффективного количества иммунореактивных клеток, содержащих антигенраспознающий рецептор, который связывает выбранный антиген, где связывание активирует иммунореактивную клетку, тем самым осуществляя лечение или предупреждение заболевания (такого как новообразование, инфицирование патогеном, аутоиммунное нарушение или реакция против аллогенного трансплантата). Дозирование средств терапии на основе CAR T-клеток может вовлекать, например, введение от 106 до 109 клеток/кг, с курсом лимфодеплеции, например циклофосфамидом, или без него.
[0590] Для защиты против возможных неблагоприятных реакций, модифицированные иммунореактивные клетки могут быть снабжены трансгенным переключателем безопасности в форме трансгена, который делает клетки чувствительными к действию определенного сигнала. Например, для этого можно использовать ген тимидинкиназы (TK) вируса простого герпеса, например, путем введения в аллогенные T-лимфоциты, используемые в качестве инфузий донорских лимфоцитов после трансплантации стволовых клеток. В таких клетках введение нуклеозидного пролекарства, такого как ганцикловир или ацикловир, вызывает клеточную смерть. Альтернативные конструкции переключателя безопасности включают индуцибельную каспазу 9, например, запускаемую посредством введения низкомолекулярного димеризатора, который сводит вместе две нефункциональных молекулы icasp9 с образованием активного фермента. Описано широкое множество альтернативных подходов для осуществления контроля клеточной пролиферации (см., например, публикацию патента США № 20130071414; публикацию патента PCT WO2011146862; публикацию патента PCT WO201401 1987; публикацию патента PCT WO2013040371). При дальнейшей доработке адоптивных способов терапии может использоваться геномное редактирование для адаптации иммунореактивнвых клеток для альтернативных вариантов применения, например, предоставление отредактированных CAR T-клеток.
[0591] Способы клеточной терапии также могут вовлекать активацию ex vivo и экспансию T-клеток. В некоторых вариантах осуществления T-клетки можно активировать до введения их индивидууму, нуждающемуся в этом. Примеры этих типов способов лечения включают использование инфильтрирующих опухоль лимфоцитов (TIL) (см. патент США №5126132), цитотоксических T-клеток (см. патент США №6255073; и патент США №5846827), подвергнутых экспансии клеток дренирующих опухоль лимфатических узлов (см. патент США №6251385) и различных других препаратов лимфоцитов (см. патент США №6194207; патент США №5443983; патент США №6040177; и патент США №5766920).
[0592] Активированная ex vivo популяция T-клеток может быть в состоянии, которое в максимальной степени организует иммунный ответ на злокачественную опухоль, инфекционные заболевания или другие болезненные состояния, например, аутоиммунное заболевание. Для активации по меньшей мере два сигнала могут быть доставлены в T-клетки. Первый сигнал обычно доставляется через T-клеточный рецептор (TCR) на поверхности T-клеток. Первый сигнал TCR обычно запускается при взаимодействии TCR с пептидными антигенами, экспрессируемыми совместно с комплексом MHC на поверхности антигенпредставляющой клетки (APC). Второй сигнал обычно доставляется через костимулирующие рецепторы на поверхности T-клеток. Костимулирующие рецепторы, как правило, запускаются соответствующими лигандами или цитокинами, экспрессируемыми на поверхности APC.
[0593] Предусматривается, что T-клетки, специфичные к иммуногенным антигенным пептидам, идентифицированным с использованием способа, описанного в настоящем описании, можно получать и использовать в способах лечения или предупреждения заболевания. В связи с этим, изобретение относится к способу лечения или предупреждения заболевания или состояния у индивидуума, включающему введение индивидууму клеточной популяции, содержащей клетки, специфичные к иммуногенным антигенным пептидам, идентифицированным с использованием способа, описанного в настоящем описании, в количестве, эффективном для лечения или предупреждения заболевания у индивидуума. В некоторых вариантах осуществления способ лечения или предупреждения заболевания у индивидуума включает введение клеточной популяции, обогащенной реактивными в отношении заболевания T-клетками, индивидууму в количестве, эффективном для лечения или предупреждения злокачественной опухоли у млекопитающего. Клетки могут представлять собой клетки, которые являются аллогенными или аутологичными для индивидуума.
[0594] Кроме того, изобретение относится к способу индукции специфического для заболевания иммунного ответа у индивидуума, вакцинации против заболевания, лечения и/или облегчения симптома заболевания у индивидуума путем введения индивидууму антигенного пептида или вакцины.
[0595] Пептид или композицию по изобретению можно вводить в количестве, достаточном для индукции ответа CTL. Антигенный пептид или вакцинную композицию можно вводить отдельно или в комбинации с другими терапевтическими средствами. Иллюстративные терапевтические средства включают, но не ограничиваются ими, химиотерапевтическое или биотерапевтическое средство, лучевую терапию или иммунотерапию. Можно вводить любое подходящее терапевтическое средство против конкретного заболевания. Примеры химиотерапевтических и биотерапевтических средств включают, но не ограничиваются ими, альдеслейкин, алтретамин, амифостин, аспарагиназа, блеомицин, капецитабин, карбоплатин, кармустин, кладрибин, цисаприд, цисплатин, циклофосфамид, цитарабин, дакарбазин (DTIC), дактиномицин, доцетаксел, доксорубицин, дронабинол, эпоэтин альфа, этопозид, филграстим, флударабин, фторурацил, гемцитабин, гранизетрон, гидроксимочевина, идарубицин, ифосфамид, интерферон альфа, иринотекан, ланзопразол, левамизол, лейковорин, магестрол, месну, метотрексат, метоклопрамид, митомицин, митотан, митоксантрон, омепразол, ондансетрон, паклитаксел (Taxol®), пилокарпин, прохлорперазин, ритуксимаб, тамоксифен, таксол, топотекана гидрохлорид, трастузумаб, винбластин, винкристин и винорелбина тартрат. Кроме того, индивидууму можно вводить антиммуносупрессивное или иммуностимулирующее средство. Например, индивидууму, кроме того, можно вводить антитело против CTLA или антитело против PD-1 или против PD-L1.
[0596] Количество каждого пептида, подлежащего включению в вакцинную композицию, и режим дозирования могут быть определены специалистом в данной области. Например, пептид или его вариант можно получать для внутривенной (в/в) инъекции, подкожной (п/к) инъекции, внутрикожной (в/к) инъекции, внутрибрюшинной (в/б) инъекции, внутримышечной (в/м) инъекции. Иллюстративные способы инъекции пептидов включают п/к, в/к, в/б, в/м и в/в. Иллюстративные способы инъекции ДНК включают в/к, в/м, п/к, в/б и в/в. Другие способы введения вакцинной композиции известны специалистам в данной области.
[0597] Фармацевтическая композиция может быть составлена так, чтобы выбор, количество и/или уровень пептидов, присутствующих в композиции, были специфическими для заболевания и/или специфическими для пациента. Например, точный выбор пептидов может определяться профилями экспрессии исходных белков в данной ткани, чтобы избежать побочных эффектов. Выбор может зависеть от конкретного типа заболевания, статуса заболевания, предшествующих режимов лечения, иммунного статуса пациента и HLA-гаплотипа пациента. Более того, вакцина по настоящему изобретению может содержать индивидуализированные компоненты в зависимости от персональных потребностей конкретного пациента. Примеры включают варьирование количеств пептидов в соответствии с экспрессией родственного антигена у конкретного пациента, нежелательные побочные эффекты вследствие персональной аллергии или других способов лечения, и коррекцию вторичных способов лечения после первого раунда или схемы лечения.
Компьютерные системы контроля
[0598] Настоящее изобретение относится к компьютерным системам контроля, которые запрограммированы для осуществления способов по изобретению. На фиг. 10 представлена компьютерная система (1001), которая запрограммирована или иным образом организована для обучения машинно-обучаемой модели для прогнозирования презентации пептидов HLA. Эта компьютерная система (1001) может регулировать различные аспекты настоящего изобретения, например, такие как ввод информации об аминокислотных положениях, перенос введенной информации в базы данных и получение обученного алгоритма с использованием наборов данных. Компьютерная система (1001) может представлять собой пользовательское электронное устройство или удаленную компьютерную систему. Электронное устройство может представлять собой мобильное электронное устройство.
[0599] Компьютерная система (1001) включает центральный процессорный элемент (CPU, также "процессор" и "компьютерный процессор" в настоящем описании) (1005), который может представлять собой одноядерный или многоядерный процессор, либо посредством последовательной обработки, либо посредством параллельной обработки. Компьютерная система (1001) также включат элемент или устройство памяти (1010) (например, память с произвольным доступом, односторонняя память, флэш-память), элемент хранения (1015) (например, жесткий диск), интерфейс связи (1020) (например, сетевой адаптер) для связи с одной или более другими системами и периферическими устройствами (1025), либо внешними, либо внутренними, либо и с теми, и с другими, такими как принтер, монитор, USB-накопитель и/или дисковод CD-ROM. Память (1010), элемент хранения (1015), интерфейс (1020) и периферические устройства (1025) соединены с CPU (1005) через соединительную шину (сплошные линии), такую как материнская плата. Элемент хранения (1015) может представлять собой элемент хранения данных (или хранилище данных) для хранения данных. Компьютерная система (1001) может быть функционально связана с компьютерной сетью ("сеть") (1030) посредством интерфейса связи (1020). Сеть (1030) может представлять собой Интернет, интернет и/или экстранет, или интранет и/или экстранет, которые сообщаются с Интернетом. Сесть (1030) в некоторых случаях представляет собой телекоммуникационную сеть и/или сеть данных. Сеть (1030) может включать один или более компьютерных серверов, которые могут обеспечивать сеть с равноправными узлами, которая поддерживает распределенные вычисления. Сеть (1030), в некоторых случаях с помощью компьютерной системы (1001), может применять структуру клиент-сервер, которая может позволять устройствам, сопряженным с компьютерной системой (1001), вести себя как клиент или сервер.
[0600] CPU (1005) может выполнять последовательность машинночитаемых инструкций, которая может быть выполнена в виде программы или программного обеспечения. Инструкции могут храниться в памяти (1010). Инструкции могут быть направлены на CPU (1005), который затем программирует или иным образом конфигурирует CPU (1005) для выполнения способов по настоящему изобретению. Примеры операций, выполняемых CPU (1005), могут включать извлечение, декодирование, выполнение и обратную запись.
[0601] CPU (1005) может быть частью контура, такого как интегральная схема. В контур может быть включен один или более других компонентов системы (1001). В некоторых случаях контру представляет собой интегральную схему специального назначения (ASIC).
[0602] Элемент хранения (1015) может хранить файлы, такие как драйверы, библиотеки и сохраненные программы. Элемент хранения (1015) может хранить пользовательские данные, например, установки пользователя или пользовательские программы. В некоторых случаях компьютерная система (1001) может включать один или более дополнительных элементов хранения данных, которые являются внешними для компьютерной системы (1001), такие как находящиеся на удаленном сервере, который сообщается с компьютерной системой (1001) через интранет или Интернет.
[0603] Компьютерная система (1001) может сообщаться с одной или более удаленными компьютерными системами через сеть (1030). Например, компьютерная система (1001) может сообщаться с удаленной компьютерной системой или пользователем. Примеры удаленных компьютерных систем включают персональные компьютеры (например, портативный PC), тонкие или планшетные PC (например, Apple® iPad, Samsung® Galaxy Tab), телефоны, смартфоны (например, Apple® iPhone, устройство на базе Android, Blackberry®) или персональные цифровые помощники. Пользователь может получить доступ к компьютерной системе (1001) через сеть (1030).
[0604] Способы, как описано в настоящем описании, могут быть выполнены посредством машинно-исполнимого кода (например, посредством компьютерного процессора), хранящегося на электронном электронной ячейке памяти компьютерной системы (1001), например, такой как внутренняя память (1010) или устройство для хранения данных (1015). Машинно-исполнимый или машинно-читаемый код может быть предоставлен в форме программного обеспечения. В ходе применения код может выполняться процессором (1005). В некоторых случаях код может извлекаться из устройства хранения (1015) и храниться в памяти (1010) для свободного доступа процессором (1005). В некоторых ситуациях устройство хранения (1015) может отсутствовать, и выполняемые инструкции хранятся в памяти (1010).
[0605] Код может быть предварительно составлен или сконфигурирован для применения с машиной, имеющей процессор, адаптированный для выполнения кода, или он может быть составлен в ходе выполнения. Код может быть предоставлен на языке программирования, который может быть выбран так, чтобы позволить выполнение кода, составленного в предварительно или в ходе выполнения.
[0606] Аспекты систем и способов, описанных в настоящем описании, такие как компьютерная система (1001), могут быть осуществлены посредством программирования. Различные аспекты технологии могут быть задуманы в качестве "продуктов" или "изделий", главным образом, в форме машинно-исполнимого кода (или исполнимого процессором) и/или ассоциированных с ним данных, которые находятся на или воплощены в виде типа машинно-читаемого носителя. Машинно-исполнимый код может храниться на устройстве хранения, таком как жесткий диск или внутренняя память (например, память с произвольным доступом, односторонняя память, флэш-память). Носитель типа "хранения" может включать любую или всю из материальной памяти компьютеров, процессоров и т.п., или ассоциированных с ними модулей, таких как различные полупроводниковая память, ленточное устройство, дисководы и т.п., которые могут обеспечить долговременное хранение в любой момент программирования программного обеспечения. Все или части программного обеспечения иногда могут сообщаться через Интернет или различные телекоммуникационные сети. Такое сообщение, например, может позволить загрузку программного обеспечения из одного компьютера или процессора в другой, например, из сервера управления или хост-компьютера в компьютерную платформу сервера приложений. Таким образом, другой тип носителя, который может включать элементы программного обеспечения, включает оптические, электрические и электромагнитные волны, такие как используются физическими интерфейсами между локальными устройствами, через проводные и оптические наземные сети и через различные пути воздушного сообщения. Физические элементы, которые несут такие волны, такие как проводные или беспроводные линии связи, оптические линии связи и т.п., также могут считаться носителем, включающим программное обеспечение. Как используют в рамках изобретения, если не ограничены долговременным материальным носителем для "хранения", термины, такие как компьютерно- или машинно-"считываемый носитель" относятся к любому носителю, который участвует в предоставлении инструкций процессору для выполнения.
[0607] Таким образом, машинно-считываемый носитель, такой как компьютерно-исполнимый код, может принимать множество форм, включая, но не ограничиваясь ими, материальный носитель для хранения, носитель на несущих частотах или физический транспортный носитель. Энергонезависимое запоминающее устройство включает, например, оптические или магнитные диски, такие как любые из устройств хранения в любом компьютере(ах) и т.п., такие как могут использоваться для исполнения баз данных, и т.д. представленных на чертежах. Энергозависимое запоминающее устройство включает динамическую память, такую как главная память такой компьютерной платформы. Материальные передающие носители включают коаксильные кабели; медную проволоку и волоконную оптику, включая провода, которые включают шину в компьютерной системе. Передающий носитель на несущих частотах может иметь форму электрических или электромагнитных сигналов, или звуковых или световых волн, таких как волны, сгенерированные в ходе радиочастотной (RF) и инфракрасной (IR) передачи данных. Таким образом, распространенные формы компьютерно-читаемого носителя включают, например: дискету, гибкий диск, жесткий диск, магнитную ленту, любой другой магнитный носитель, CD-ROM, DVD или DVD-ROM, любой другой оптический носитель, бумажная лента перфорированной карты, любой другой физический носитель для хранения с паттернами отверстий, RAM, ROM, PROM и EPROM, FLASH-EPROM, любой другой чип или кассету памяти, передающие данные и инструкции на несущих частотах, кабели или соединения, передающие такую несущую частоту, или любой другой носитель, с которого компьютер может считывать программирующий код и/или данные. Многие из этих форм компьютерно-читаемых носителей могут быть вовлечены в передачу одной или более последовательностей одной или более инструкций на процессор для выполнения.
[0608] Компьютерная система (1001) может включать или может сообщаться с электронным дисплеем (1035), который включает пользовательский интерфейс (UI) (1040) для предоставления, например, вероятности того, что один или более белков, кодируемых аллелем MHC класса II злокачественной клетки индивидуума, будет презентировать данную последовательность идентифицированных пептидных последовательностей. Примеры UI включают, но не ограничиваются ими, графический пользовательский интерфейс (GUI) и пользовательский интерфейс в сетевом исполнении.
[0609] Способы и системы по настоящему изобретению могут быть осуществлены посредством одного или более алгоритмов. Алгоритм может быть осуществлен посредством программного обеспечения при выполнении центральным процессорным элементом (1005). Алгоритм, например, может вводить информацию об аминокислотных положениях, передавать введенную информацию в наборы данных и генерировать обученный алгоритм с использованием наборов данных.
ПРИМЕРЫ
[0610] Примеры, приведенные ниже, приведены только для иллюстративных целей и не ограничивают объем формулы изобретения, приведенной в настоящем описании.
ПРИМЕР 1. Эффективность прогнозирования связывания HLA класса II
[0611] В этом примере использовали валидирующий набор данных, включающий выявленные посредством масс-спектрометрии пептиды и пептиды-ловушки, полученные в соотношении 1:19 (наилучшие последовательности:ловушки) посредством случайной перестановки наиболее подходящих пептидов, для анализа эффективности средства прогнозирования связывания neonmhc2 (NEON) и NetMHCIIpan (фиг. 4). Для средства прогнозирования NEON конструировали отдельную модель для каждого представленного аллеля MHC II. Высота столбцов продемонстрировала положительную прогностическую величину (PPV). Аллели отсортированы по эффективности модели при прогнозировании для этого аллеля. Средство прогноза связывания NEON продемонстрировало более высокую PPV для всех аллелей по сравнению с NetMHCIIpan.
[0612] В этом примере также тестировали эффект пороговых значений SPI на валидацию средства прогнозирования связывания (фиг. 5). Эффективность средства прогнозирования связывания HLA класса II показана после обучения/валидации на наборах пептидов с различными предельными значениями оцененной интенсивности пика (SPI). Использовали различные предельные значения для SPI: обучение и оценка на наборах данных с использованием наблюдаемых в MS наилучших пептидов с SPI более чем или ровно 70, обучение на пептидах с SPI более чем или ровно 50 и валидация на пептидах с SPI более чем или ровно 70, и обучение и валидация на пептидах с SPI более чем или ровно 50.
[0613] В этом примере собраны данные для 35 аллелей HLA-DR, которые имеют популяционный охват >95% для HLA-DR (частоты аллелей в США), чтобы продемонстрировать количество наблюдаемых пептидов посредством определения аллельного профиля посредством LC-MS/MS с предельными значениями оцененной интенсивности пика (SPI) более или ровно 70 (фиг. 6).
[0614] В одном иллюстративном случае применяли модельный анализ для тестовой части данных для указанных аллелей HLA класса II, которые получены на настоящий момент для программы Neonmhc2. Тестовая часть данных состояла из положительного примера (например, пептид из выборки наиболее подходящих пептидов), которые представляют собой связывающиеся с классом II структуры, выявленные посредством MS, и отрицательных примеров (например, пептид из выборки ловушек), которые представляют собой версии положительных примеров, полученные посредством перестановки. Соотношение наиболее подходящая последовательность: ловушка составляло 1:19, например, для каждого положительного образца было включено 19 отрицательных образцов (т.е. положительная на 5% выборка) и для валидации использовали тестовую часть. Показатели PPV обобщали посредством отбора 5% пептидов с наилучшей оценкой в тестовой части и установления, какая доля из них являются положительными. Результаты указаны на фиг. 7A.
[0615] Наблюдалось, что для полученных аллелей HLA-DR, когда размер обучающей выборки возрастал, величина PPV возрастала (фиг. 7B).
[0616] В этом примере связанные с процессингом переменные далее улучшали прогнозирование (фиг. 8). В обучающей части данных проводили аппроксимацию логистической регрессии для прогнозирования презентации HLA класса II с использованием силы связывания (средство прогноза NetMHCIIpan или Neon) и признаков процессинга (экспрессия согласно RNA-Seq и установленная составляющая смещения для генного уровня). При отдельном оценочном распределении положения экзонов, перекрывающие выявленные при MS пептиды MHC II ("наиболее подходящие последовательности") могут быть оценены вдоль случайных экзонных положений, не выявленных при MS (соотношение 1:499). Главным образом, Neon со связанными с процессингом переменными продемонстрировал более высокую PPV, чем NetMHCIIpan, средство прогнозирования Neon, и NetMHCIIpan со связанными с процессингом переменными.
ПРИМЕР 2. Архитектура нейронной сети
[0617] В этом примере нейроннную сеть использовали для получения обучающего алгоритма (фиг. 9). Вводимые пептиды представлены в качестве 20-меров, причем более короткие пептиды дополнены символами "отсутствия". Каждый пептид имел 31-мерное вложение, так что входные данные в нейронную сеть представляли собой матрицу 20×31. Перед обработкой нейронной сетью проводилась нормализация признаков на матрице 20×31 на основе средних значений величин признаков и стандартных отклонений в обучающей выборке. Первый сверточный слой имел ядро из 9 аминокислот и 50 фильтров (также называемых каналами) с активационной функцией ReLU. После этого следовала пакетная нормализация, затем пространственное исключение с частотой исключения 20%. После этого следовал другой сверточный слой с ядром из 3 аминокислот и 20 фильтрами с активационной функцией ReLU, за которыми вновь следовала пакетная нормализация и пространственное исключение с частотой исключения 20%. Затем применяли глобальный максимальный пулинг, учитывая максимально активированный нейрон в каждом из 20 фильтров; затем эти 20 величин передаются в полносвяазнный (плотный) слой с единичным нейроном с использованием сигмовидной активационной функции. Результат этого слоя обрабатывали в качестве прогноза связывание/не связывание. Регуляризацию L2 применяли для веса первого сверточного слоя, второго сверточного слоя и плотного слоя с весом 0,05, 0,1 и 0,01, соответственно.
ПРИМЕР 3. Масштабируемый протокол моноаллельного определения профиля лигандов MHC класса II
[0618] В настоящее время знание о связывающих мотивах MHC класса II может быть основано на двух анализах связывания in vitro, один из которых вычисляет EC50 с использованием клеточного MHC, а другой из которых вычисляет IC50 с использованием очищенного MHC. Основной алгоритм прогнозирования HLA класса NetMHCIIpan обучен исключительно на этих данных.
[0619] Ограниченное количество аллелей HLA класса II человека в настоящее время подтверждается более чем 200 примерами подтвержденных связывающих пептидов (аффинность <100 нМ) (фиг. 12E), практически все представляют собой 15-меры. Эти эксперименты охватывают только наиболее распространенные аллели HLA-DR у европиоидов с ограниченным охватом аллелей, специфических для не европиоидных популяций (например, HLA-DRB1*15:02) и практически отсутствием охвата распространенных аллелей HLA-DP и HLA-DQ. Современная эффективность прогнозирования для HLA класса II, даже на распространенных аллелях европиоидов, значительно отстает от MHC класса I; кривые ROC только умерено лучше, чем случайные.
[0620] Учитывая эти ограничения, в рамках настоящего изобретения была разработана новая биотехнология, которая была названа моноаллельным улавливанием посредством улавливания меченых аллелей (MAPTAC™), которая позволяет эффективное выделение связывающих HLA класса II пептидов, которые связываются с белком MHC, кодируемым единичным аллелем, для идентификации на основе MS (фиг. 11A и 11B); этот подход также применим для HLA класса I. Как используют для HLA класса II, цепи альфа и бета выбранного аллеля кодируют на генетической конструкции с последовательностью биотин-акцепторного пептида (BAP), помещенной на C-конце бета-цепи. Затем эти клетки лизируют и инкубируют с ферментом BirA для биотинилирования C-конца бета-цепи улавливающего аллеля. Соосаждение с нейтравидином очищает популяцию связанных с MHC пептидов, которые далее выделяют посредством эксклюзионной хроматографии и секвенируют с использованием наилучших в своем классе протоколов LC-MS/MS.
[0621] В некоторых вариантах осуществления анализ LC-MS/MS оценивают с использованием спектрометрии подвижности ионов в несимметричном поле высокой напряженности (FAIMS). В некоторых вариантах осуществления пептиды подвергают как кислотному обращенно-фазовому (aRP), так и основному обращенно-фазовому (bRP) фракционированию перед анализом посредством nLC-MS/MS.
[0622] Двухдневная трансфекция была достаточной для достижения устойчивой экспрессии конструкции (фиг. 12B) с соответствующей локализацией на клеточной поверхности в трех различных клеточных линиях (expi293, A375 и B721) для четырех различных аллелей (фиг. 12C).
[0623] Поскольку HLA-DRA функционально инвариантен, этот подход достигает одноаллельного разрешения, даже если улавливающие бета-цепи образуют пару с эндогенной альфа-цепью. Это означает, что этот подход может использоваться для определения профиля аллелей HLA-DR независимо от предсуществующего генотипа HLA и уровня экспрессии в данной клеточной линии.
[0624] Для HLA-DP и HLA-DQ, как альфа-, так и бета-цепи являются переменными, и обе из них участвуют в связывании пептидов, так что одноаллельное разрешение может ожидаться, только если нативный аллель является гомозиготным и соответствует улавливающему аллелю. Альтернативно можно использовать улавливание только посредством бета-цепи для установления общей информации о пептидах, соответствующих нативной альфа-цепи.
[0625] Подвергнутые определению профиля аллели включали пять аллелей HLA-DR (DRB1*03:01, DRB1*09:01, DRB1*11:01, DRB3*01:01 и DRB3*02:02), а также один аллель HLA-DP (DPB1*01:01/DPA1*01:03), один аллель HLA-DQ (DQB1*06:02/DQA1*01:02) и два аллеля класса I (таблица 1). Во всех случаях 2-3 повторения было достаточно для наблюдения по меньшей мере 1500 уникальных пептидов (фиг. 11B). Среди подвергнутых определению профиля аллелей только небольшой процент наиболее подходящих последовательностей соответствовал известным примесям или полностью трипсинизированным последовательностям; с другой стороны, имитирующие трансфекции обеспечивали относительно мало пептидов, которые по большей части идентифицировались как известные примеси или полностью трипсинизированные последовательности (фиг. 11B).
В таблице 1 представлено обобщение образцов, использованных в иллюстративных экспериментах.
Таблица 1
[0626] Поскольку концы связывающих MHC II пептидов не должны вмещаться в связывающую бороздку MHC, множество различных типов структур могут связываться в равной степени хорошо, если они имеют общую центральную связывающую последовательность. Когда пептиды объединяли с перекрывающейся последовательностью во "вложенные множества", наблюдали 500-700 уникальных вложенных множеств на аллель HLA класса II; ни, как правило, происходили из 500-600 уникальных генов. Распределения длин для пептидов, связывающих HLA класса I и HLA класса II, соответствуют распределениям длин, наблюдаемым в предшествующих исследованиях на основе MS, в которых использовалось соосаждение на основе антител (фиг. 11C).
[0627] Среди предполагаемых MHC-связывающих пептидов большинство аминокислот было представлено на уровнях, согласующихся с их исходными протеомными частотами. Исключения включали цистеин, метионин и триптофан, уровень которых был уменьшен, что согласовывалось с предшествующими исследованиями на основе MS для пептидов MHC II. Уменьшение представленности цистеина, метионина и триптофана не наблюдалось в совпадающих по аллелю высокаффинных пептидах (<50 нМ) из IEDB; однако пептиды IEDB демонстрировали увеличение уровня лейцина и метионина и уменьшение уровня пролина, аспарагиновой кислоты и глутаминовой кислоты в отношении протеома.
ПРИМЕР 4. Протокол MAPTAC™ раскрывает известные и новые связывающие MHC II мотивы
[0628] Поскольку связывающая MHC подпоследовательность пептидов класса II не находится в фиксированном положении относительно N- или C-конца, точное выявление мотивов класса II должно динамически учитывать различные возможности уровня связывания для каждого связывающего пептида. Инструмент Gibb's Cluster tool направлен на эту проблему посредством алгоритма ожидания-максимизации (EM). Проводилось изучение применения подхода обнаружения новых мотивов с использованием сверточных нейронных сетей (CNN). CNN были успешными в области компьютерного зрения, которое аналогично направлено на достижение трансляционно инвариантного распознавания паттернов. CNN были обучены для отличения связывающих MHC пептидов от их перемешанных версий, а затем выравнивания положительных примеров в соответствии с подпоследовательностями, которые достигли максимальной активации узлов в предпоследнем слое сети. Как применялось к моноаллельным данным MS, этот подход обеспечивал мотивы, согласующиеся с кластеризацией Gibb, и продемонстрировал якоря в соответствующих положениях 1, 4, 6 и 9 (фиг. 13). Эти мотивы в высокой степени согласовывались с происходящими из CNN мотивами, наблюдаемыми для высокоаффинных связывающих структур из IEDB (аффинность <50 нМ; фиг. 13). Для DRB1*11:01 было далее подтверждено, что мотив был стабильным среди клеточных линий и соответствовал гомозиготной по DRB1*11:01 клеточной линии, для которой ранее был определен профиль с использованием общего антитела против DR. Аналогично, мотивы, полученные для аллелей MHC класса I, согласовывались с мотивами из способов на основе аффинности и предшествующих исследований на основе MS (фиг. 14A).
[0629] Хотя все аллели MHC класса II продемонстрировали характерные мотивы, энтропия в якорных положениях была заметно более высокой, чем наблюдалось для аллелей MHC класса I. Таким образом, были определены предпочтительные аминокислоты в каждом якорном положении для каждого аллеля MHC класса II, и было обнаружено, что только 10-20% пептидов демонстрируют идеальные остатки во всех четырех якорных положениях и вплоть до 60% демонстрируют два или менее ожидаемых якорей (фиг. 14B и фиг. 30C). 13-17-меры оценивали в отношении потенциала связывания с использованием NetMHCIIpan, и, в то время как выявленные посредством MS пептиды были имели более высокий спрогнозированный связывающий потенциал во всех случаях, существовало значительное перекрывание с показателями совпадающих по длине случайных пептидов (фиг. 14C, и фиг. 36A).
ПРИМЕР 5. Алгоритмы, обученные на данных моноаллельной MS для MHC II, прогнозируют иммуногенность
[0630] Далее, рассматривали, могут ли данные из моноаллельной платформы MS обеспечивать улучшенные прогнозы связывания MHC класса II. Основываясь на подходе CNN, создавали многослойную сеть с размерами фильтров, скачкообразными соединениями и общим рецептивным полем (фиг. 31A). Для обучения и оценки этой модели глубокого обучения, названной neonmhc2, протеом распределяли на три части, соответствующих 75%, 12,5% и 12,5% генов. Первую часть использовали для обучения CNN посредством стохастического градиентного спуска, и вторую использовали для оптимизации архитектуры и оптимизации гиперпараметров. Третью часть использовали для оценки эффективности только один раз в конце анализа. Чтобы гарантировать целостность оценки, обращали внимание на то, чтобы все гены в паралогичных группах генов были помещены в одну часть.
[0631] Поскольку MS демонстрирует некоторую степень смещения в отношении остатков, в частности, в отношении цистеина (фиг. 12D), эту проблему уменьшали посредством использования негативных обучающих примеров (называемых ловушками), полученных посредством случайной перестановки последовательностей положительных примеров. Поскольку этот подход несет в себе риск выучивания свойств последовательностей натуральных белков, что может искусственно увеличивать эффективность прогнозирования, модельная оценка использовала отличающуюся стратегию получения ловушек, где ловушки отбирали случайным образом из не выявленных подпоследовательностей генов-источников пептидов. Вычисление положительной прогностической величины (PPV) в соотношении наиболее подходящей последовательности и ловушки 1:19 продемонстрировало, что neonmhc2 имеет улучшенную PPV относительно NetMHCIIpan при прогнозировании пептидов посредством MS в оценочной части (фиг. 4 и фиг. 31B). Эксперименты, в которых искусственным образом снижался размер обучающей выборки данных neonmhc2, указывают на то, что ее эффективность ограничивается данными и может улучшаться при более глубоком охвате данных (фиг. 16).
[0632] Исследовали способность neonmhc2 прогнозировать аффинность связывания - тип данных, по которым обучена NetMHCIIpan. Чтобы лишить NetMHCpan преимущества обучения и оценки на одних и тех же показателях пептидов, оценку проводили с использованием несколько более старой версии NetMHCIIpan, оценивающей пептиды, депонированные в IEDB. С использованием тау-статистики Кендалла для оценки точности прогнозирования, NetMHCIIpan осуществляет оценку аналогично или более лучше, чем средство прогнозирования на основе MS во всех случаях (фиг. 15B). Интересно, что эффективность зависела от типа проведенного анализа аффинности. В то время как neonmhc2 умеренно отставало от NetMHCIIpan при прогнозировании показателей аффинности, осуществленном Sette и коллегами, оно более значительно отставало от NetMHCIIpan при прогнозировании показателей, осуществленном Buus и коллегами. Учитывая эти результаты в совокупности, оказалось, что между этими платформами существуют присущие им отличия, однако не было непосредственно очевидно, какой из этих подходов был более точным.
[0633] Для достижения большей ясности оценивали способность прогнозировать ответы натуральных CD4 T-клеток. Данные IEDB были в основном непригодными для этой цели, поскольку аллельная рестрикция ответов практически всегда является либо неопределенной, либо расчетной. Таким образом, проводили сборку большого набора данных направляемого тетрамером картирования эпитопов (TGEM). Во всех этих исследованиях использовался полный перекрывающийся скрининг пептидов вместо приоритезации прогнозирования, устраняющий смещение наблюдения в пользу NetMHCIIpan. Между тем, аллельная рестрикция является недвусмысленной. Для всех аллелей, для которых существовало достаточно данных для оценки, neonmhc2 существенно превосходило NetMHCpan, которое функционировало только несколько лучше, чем случайным образом. Таким образом, платформа MAPTAC™ может быть лучшей в своем классе для обучения моделей, которые идентифицируют иммуногенные эпитопы MHC класса II.
ПРИМЕР 6. Алгоритмы, обученные на мультиаллельных данных MS, являются худшими
[0634] Учитывая, что существуют многочисленные мультиаллельные базы данных класса II, доступные общественности, на основе стандартной очистки с использованием общих антител против DR и общих антител против II проводили тестирование того, может ли подходящее средство прогнозирования быть обучено с использованием только мультиаллельных данных. Несколько групп продемонстрировали успех обратной свертки мотивов аллелей MHC класса I из мультиаллельных данных класса I, хотя эти усилия еще не были транслированы в общедоступное средство прогнозирования. Кроме того, обратная свертка мотивов класса II осложнена необходимостью одновременно устанавливать как уровень связывания, так и кластерную принадлежность каждого пептида. В то время как инструмент Gibbs Cluster tool используется для исследования возможности обратной свертки для класса II, точность этого подхода еще не подтверждена тщательно.
[0635] Для оценки точности обратной свертки для класса II были выбраны общедоступные базы данных общих DR с известным генотипом. Для каждого набора данных двадцать пептидов из моноаллельных данных, полученных авторами изобретения, вносили для каждого аллеля в донорском генотипе (1-2 аллеля DR1 плюс 0-2 аллеля DR3/4/5, в зависимости от гаплотипа и зиготности). Инструмент Gibbs Clustering tool применяли для каждого набора данных, и то, подвергались ли внесенные пептиды надлежащей сокластеризации, наблюдали в соответствии с их известным аллелем-источником. В ранних версиях этого анализа, либо количество кластеров для номера аллеля был фиксированным, либо кластеру Gibbs позволяли автоматически определять наиболее оптимальное количество кластеров; однако оказалось, что ни один из подходов не осуществлял обратную свертку пептидов в точности. Чтобы помочь алгоритму, наиболее оптимальное количество кластеров выбирали путем вычисления скорректированной взаимной информации между аллелями-источниками внесенных пептидов и присвоенными им кластерами. Тем не менее, во всех кроме нескольких случаев, пептиды распределялись между кластерами независимо от их аллеля-источника (фиг. 17A). Эти результаты указывают на то, что современные протоколы обратной свертки могут не быть достаточно точными для MHC класса II.
[0636] Одним из ограничений этого анализа является то, что некоторые пептиды могут быть способны связывать более одного аллеля. В соответствии с этим, следующий вопрос состоит в том, могут ли связывающие мотивы, полученные из мультиаллельных данных, тем не менее, соответствовать мотивам, выявленным из моноаллельных данных. Для оценки этого кластеры с наилучшим соответствием с улавливающими пептидами для каждого единичного аллеля отбирали и конструировали мотивы на основе этих популяций (см., например, фиг. 17B). В то время как многие мотивы отчетливо продемонстрировали некоторые из известных якорей, другие положения были дискордантными с моноаллельным мотивом или дискордантными между наборами исходных данных. Кроме того, были явные случая, когда появлялись ложные якоря. Наконец, авторы изобретения оценили, можно ли подвергнутые обратной свертке данные использовать для обучения CNN, которые могут прогнозировать в оценочной части их моноаллельного набора данных. Модели, обученные на подвергнутых обратной свертке мультиаллельных данных, во всех случаях были хуже обученных посредством MAPTAC™ моделей (фиг. 17C).
ПРИМЕР 7. Признаки исходного белка влияют на вероятность презентации
[0637] Для MHC класса I протеасома играет важную роль в определении репертуара презентируемых эпитопов; таким образом, проводили охарактеризацию того, как процессинг белка в пептид определяет репертуар класса II.
[0638] Сначала фокусировались на точных положениях N- и C-концов пептидов MHC класса II, наблюдаемых в нескольких наборах данных, полученных посредством определения профиля пептидов на основе тканей. При сравнении частот аминокислот по положениям относительно пептидов-ловушек наблюдали значительное увеличение и снижение уровней. Этот паттерн согласуется с недавними наблюдениями. Интересно, что общий паттерн не соответствует известному предпочтительному для расщепления паттерну катепсина S ([RPI][FMLW][KQTR][ALS]), наиболее охарактеризованному ферменту процессинга для класса II.
[0639] Для определения прогностического потенциала этого мотива создавали средство прогнозирования на основе NN для N- и C-концов и логистическую регрессию, которая использовала две переменных расщепления вместе со спрогнозированным потенциалом к связыванию (согласно обученной посредством MS CNN), подвергали аппроксимации для отличения истинных пептидов MS от совпадающих по длине пептидов-ловушек, отобранных из тех же генов-источников.
[0640] Это средство прогнозирования обеспечивало умеренное улучшение в отношении прогнозирования пептидов относительно модели, которая учитывала только потенциал к связыванию; однако поскольку иммуногенность связывающих MHC класса II эпитопов (взаимозаменяемо называемых эпитопами класса II) может не зависеть от точного положения расщепления пептида, вопрос состоит в том, будет ли модель все еще увеличивать величину, если точный участок расщепления неизвестен. Таким образом, схему прогнозирования прогоняли второй раз, скрывая точные положения расщепления наиболее подходящих последовательностей и ловушек, вместе оценки составных показателей способности к расщеплению среди белковых положений вблизи (+/-15 AA) введенного центра связывания. Интересно, что отсутствовало улучшение эффективности относительно средства прогнозирования только с учетом связывания. Эти результаты согласуются с предшествующей работой, которая показала, что добавление прогноза расщепления для класса II может улучшить прогнозирование для выявленных посредством MS лигандов, но не для распознавания T-клетками, которое предположительно инвариантно для конкретных концов пептидов.
[0641] Была предложена модель, в которой значительная часть пептидов MHC II "пережевывалась обратно" с их N- и C-концов после связывания MHC. Согласно этой модели, предпоследняя сигнатура пролина появляется, поскольку пролин блокирует процессинг экзопептидазами. В этом сценарии мотив, полученный посредством прямого анализа концов лигандов MHC, потенциально является некорректным, поскольку он отражает последующее редактирование вместо первоначальной стадии образования пептидных фрагментов. Таким образом, вблизи пептидов класса II были определены другие признаки последовательностей, которые могут быть способными объяснить их образование. Во-первых, проводили поиск канонической сигнатуры катепсина S, однако отсутствовало увеличение количества участков катепсина S вблизи выявленных посредством MS пептидов класса II против совпадающих по длине пептидов-ловушек, взятых из генов-источников пептидов. Поскольку эта сигнатура процессинга может отражать комплексную сборку ферментов, CNN de novo была обучена на основе вышерасположенного и нижерасположенного белкового контекста (±25 AA) для выявленных пептидов и ловушек.
[0642] Рассматривали третью модель, в которой доступность пептида определяют по свернутому или полусвернутому состоянию белка вместо его первичной последовательности. ACCPRO на основе гомологии использовали для прогнозирования вторичной структуры и областей доступности растворителя, и совокупность прогностических параметров использовали для идентификации внутренне разупорядоченных доменов.
[0643] Если предпочтительные области процессинга по своей природе трудно спрогнозировать, может быть возможным создание каталога всех белковых областей, охватываемых по меньшей мере одним пептидом в большой коллекции ранее опубликованных данных мультиаллельной MS для класса II, и использование перекрывания в качестве прогностического признака. По общему признанию, признак перекрывания искажается посредством информации о связывании, поскольку аллели, представленные в ранее опуликованных данных, могут иметь те же или сходные связывающие мотивы. Тем не менее даже этот признак только умеренно улучшал прогнозирование презентируемых пептидов, что указывает на то, что пептиды MHC класса II могут не быть сильными горячими точками процессинга.
[0644] Следующим вопросом было, какие гены в наибольшей степени вносят вклад в репертуар связывающих пептидов класса II. Уже известно, что признаки генного уровня, такие как уровень экспрессии, обеспечивают значительное усиление при прогнозировании лигандов MHC класса I. Используя ранее опубликованные наборов данных MS, определяющие профиль репертуаров связывания для класса II в тканях человека, было обнаружено, что выявленные посредством MS пептиды экспрессируются на более высоком уровне, чем случайные пептиды-ловушки (взятые из протеома), на порядок величины (фиг. 18A). Тем не менее было отмечено, что приблизительно 5% пептидов класса II картируются на гене, который по-видимому не экспрессируется в соответствии с репрезентативными данными RNA-Seq. Исходя из этого паттерна, степень, с который каждый ген был представлен на повышенном или сниженном уровне в репертуаре пептидов класса II, пытались количественно определить путем предложения базового ожидания, что количество наблюдений для каждого гена должно быть пропорционально результату умножения его длины и уровня экспрессии (фиг. 18B). Среди генов, представленных на повышенном уровне, очевидно были на более высоком уровне представлены белки, экспрессируемые в тканевой жидкости человека, в которой продуцировалось множество связывающих класс II пептидов, но, по-видимому, не экспрессируемые в нативной ткани. Это согласуется с известной ролью MHC класса II в презентации антигенов, полученных из внеклеточной среды.
[0645] Поскольку аутофагия является другим общепризнанным каскадом процессинга для класса II, определяли соотношение выявленных и ожидаемых пептидов для каждого гена (исключая любой ген с менее чем пятью выявленными пептидами и менее чем пятью ожидаемыми пептидами), и определяли, происходило ли обогащение в отношении физических партнеров известных генов аутофагии или генов, стабилизированных посредством нокаута Atg5 у мышей (фиг. 18C). Оказалось, что ни один из наборов генов не был представлен на увеличенном уровне в данных для класса II; в действительности, физические партнеры генов аутофагии, по-видимому, были представлены в несколько меньшей степени.
[0646] Изучая все клеточные локализации (фиг. 18D и фиг. 18E), несколько компартментов были определенно представлены в большей или меньшей степени. Двумя из наиболее представленных компартментов были клеточная мембрана и лизосома, каждая из которых генерировала количество пептидов для класса II, приблизительно в два раза превышающее ожидаемое количество. Неясно, связана ли повышенная представленность мембранных белков с рециклированием мембраны в аутофагосомы или аппарат Гольджи, направляющие мембранные белки непосредственно на путь аутофагии. Кажущееся противоречие между увеличенной представленностью лизосомальных белков и ранее наблюдаемой сниженной представленностью генов аутофагии показало, что эти тенденции являются в высокой степени чувствительными к конкретной подгруппе рассматриваемых связанных с аутофагией генов. На фиг. 18F представлена относительная согласованность выявления пептидов в отношении двух различных профилей экспрессии генов: в массивной опухоли и профессиональных антигенпредставляющих клетках.
ПРИМЕР 8. Точное прогнозирование MHC II требует понимания каскада эндоцитоза
[0647] В дополнение к пониманию исходного каскада для генов класса II, может быть критичным понимание того, какие типы клеток ответственны за основную часть презентации класса II. В случае злокачественной опухоли, полагают, что непрофессиональные APC, включая фибробласты и саму опухоль, презентируют класс II в воспалительном микроокружении опухоли (TME). Для дальнейшего понимания экспрессию HLA-DRB1 анализировали в трех недавно опубликованных наборах данных RNA-Seq единичных клеток, которые определяли профиль рака легкого, рака головы и шеи и меланомы. Усредняя среди клеток для уровня по типу клеток пациента, было очевидно, что канонические APC (макрофаги, дендритные клетки и B-клетки) презентируют значительно более высокие уровни класса II, чем другие типы стромальных клеток, и эта тенденция согласуется для множества пациентов и типов опухолей.
[0648] Чтобы изучить, нарушает ли иммунотерапия эту тенденцию, анализировали дополнительные данные RNA-Seq единичных клеток из отвечающих на блокаду точек контроля типов опухолей, и оценивали экспрессию HLA-DRB1 до и после лечения. Группа меланомы, которая включала одного подтвержденного отвечающего пациента, продемонстрировала единообразно низкую экспрессию HLA-DRB1 опухолевыми клетками в биоптатах как до терапии, так и после терапии (фиг. 19C). Группа базально-клеточной карциномы, которая продемонстрировала клиническую частоту ответа 55% на терапию против PD-1, аналогично продемонстрировала низкую экспрессию происходящего из опухоли HLA-DRB1 независимо от момента времени (фиг. 19C).
[0649] Эти результаты показали, что основная часть внутриопухолевой презентации HLA класса II обеспечивается в основном профессиональными APC и условия "горячего" TME не гарантируют отклонения от общего паттерна.
[0650] Поскольку опухолевые клетки могут превзойти количеством APC в микроокружении опухоли, их более низкие уровни экспрессии MHC класса II тем не менее могут быть иммунологически значимыми. Для оценки того, насколько экспрессия класса II в целом происходит из опухолевых клеток против стромы, идентифицировали пациентов TCGA с мутациями в специфических для класса II генах (фокусируясь на CIITA, CD74 и CTSS) и определяли долю чтений RNA-Seq, продемонстрировавших соматический (опухолеспецифический) вариант. Эту информацию использовали для вычисления того, какая доля экспрессии HLA-DRB1 происходила из опухоли против стромы (фиг. 19B). На основе мутаций, идентифицированных у 153 пациентов, имевших 17 различных типов опухолей, наблюдали доминантный паттерн, в соответствии с которым основная часть экспрессии II, по-видимому, происходит в неопухолевых клетках. Фокусируясь только на пациентах с высокими уровнями T-клеточной инфильтрации (наивысшие 10% при идентификации с использованием ранее опубликованной сигнатуры из 18 генов (Ayers et al., 2017)), низкая опухолевая экспрессия HLA-DR тем не менее оказалась нормой, причем только у 3 из 16 пациентов имели >1000 TPM (прогрессирование и метастазирование опухоли).
[0651] Чтобы изучить, нарушает ли иммунотерапия эту тенденцию, анализировали дополнительные данные RNA-Seq единичных клеток из отвечающих на блокаду точек контроля типов опухолей, и оценивали экспрессию HLA-DRB1 до и после лечения. Группа меланомы, которая включала одного подтвержденного отвечающего пациента, продемонстрировала единообразно низкую экспрессию HLA-DRB1 опухолевыми клетками в биоптатах как до терапии, так и после терапии (фиг. 19C). Группа базально-клеточной карциномы, которая продемонстрировала клиническую частоту ответа 55% на терапию против PD-1, аналогично продемонстрировала низкую экспрессию происходящего из опухоли HLA-DRB1 независимо от момента времени (фиг. 19C).
[0652] Эти результаты показали, что основная часть внутриопухолевой презентации HLA класса II обеспечивается в основном профессиональными APC и условия "горячего" TME не гарантируют отклонения от общего паттерна.
ПРИМЕР 9. Новые концепции прогнозирования обеспечивают более точную идентификацию иммуногенных неоантигенов
[0653] Чтобы изучить применимость neonmhc2 и ассоциированных правил процессинга, рассматривали эффективность в нескольких сценариях прогнозирования. Сначала оценивали способность к прогнозированию идентифицированных посредством MS пептидов на PMBC от семи здоровых доноров, для которых был определен профиль посредством общего антитела против DR. Этот анализ может контролировать любые системные смещения, присущие системе MAPTAC™ или продуцирующим клеточным линиям авторов настоящего изобретения. С использованием соотношения 1:499 наиболее подходящих последовательностей и ловушек и отбора ловушек случайным образом из кодирующего белки экзома проводили оценку положительной прогностической величины базовых моделей neonmhc2 и NetMHCIIpan, а также моделей, которые включали дополнительные признаки процессинга (экспрессия, смещение генного уровня согласно фиг. 18B, и перекрывание с предыдущим пептидом MHC II). Эти модели подтвердили значительное улучшение прогнозирования как связывания, так и процессинга (фиг. 20).
[0654] На фиг. 21A представлено сравнение NetMHCIIpan и neonmhc2 с дополнительным параметром процессинга или признаками, как указано. Профиль эффективности прогнозирования для восьми образцов MS определяли посредством антитела против HLA-DR (те же образцы, которые были проанализированы в примере 6, фиг. 17A). Средства прогнозирования в минимальной степени используют прогнозирование связывания HLA (либо NetMHCIIpan, либо neonmhc2) и необязательно используют дополнительные связанные с процессингом переменные: экспрессию генов, генное смещение (например, согласно фиг. 18B, фиг. 18C, фиг. 19B), и перекрывание с ранее выявленным пептидом HLA-DQ. В этом примере ловушки были отобраны из протеома случайным образом (включая гены, которые никогда не продуцировали выявленный посредством MS пептид) для достижения соотношения 1:499 наиболее подходящих последовательностей и ловушек, которое практически насыщает доступные последовательности ловушек. Положительную прогностическую величину вычисляли аналогично фиг. 4, например, наивысшие 0,2% пептидов называли положительными результатами и PPV представляет собой долю положительных результатов, которые являются истинными выявленными посредством MS пептидами. Для каждого пептида-кандидата в каждом образце показатель связывания вычисляли в качестве максимума для аллелей HLA-DR, присутствующих в генотипе образца. Хотя существует закономерная корреляция в тенденциях пептидов, выявленных посредством двух способов, модель, описанная в настоящем описании, демонстрирует более устойчивый результат. На фиг. 21B (также см. фиг. 33B) представлена эффективность прогнозирования для происходящих из опухоли пептидов, презентируемых дендритными клетками (лизат), с использованием того же соотношения наиболее подходящая последовательность: ловушка и параметров эффективности, что и на фиг. 21A. Эффективность представлена для NetMHCIIpan и моделей, описанных в настоящем описании, с использованием или без использования признаков процессинга. На фиг. 21C представлен уровень экспрессии и показатель генного смещения для каждого меченного тяжелой меткой пептида. На фиг. 21D представлена диаграмма, показывающая перекрывание генов-источников меченных тяжелой меткой пептидов в соответствии с лизатом и экспериментами по обработке УФ-излучением.
ПРИМЕР 10. Экспрессия пептидов HLA класса II в клеточных линиях и выделение MHC-II-связанных пептидов
Конструирование конструкции, клеточная культура и иммунопреципитация HLA-пептид
[0655] В этом экспериментальном исследовании получали моноаллельные клеточные линии посредством трансфекции единичной меченной аффинной меткой конструкции HLA в клеточные линии (A375, HEK293T, Expi293, HeLa), и меченные аффинной меткой комплексы HLA-пептид подвергали иммунопреципитации. На фиг. 12A и 12E, частоты аллелей MHC класса II представляют собой аллельные частоты, полученные с allelefrequencies.net/, если нет иных указаний. Аллельные частоты для популяции США вычисляли, предполагая смешение 62,3% епропиоидов, 13,3% афроамериканцев, 6,8% азиатов и 17,6% латиноамериканцев.
[0656] Что касается фиг. 12A и фиг. 12E, набор данных mhc_ligand_full.csv был загружен из данных IEDB 21 сентября 2018 года. Для валидных показателей аффинности требовалось, чтобы "способ/технология" соответствовали следующим: "клеточный MHC/конкурентный/флуоресценция", "клеточный MHC/конкурентный/радиоактивность", "клеточный MHC/прямой/флуоресценция", "очищенный MHC/конкурентный/флуоресценция", "очищенный MHC/конкурентный/радиоактивность" или "очищенный MHC/прямой/флуоресценция", и "анализируемая группа" соответствовала следующим: "константа диссоциации KD", "константа диссоциации KD (~EC50)", "константа диссоциации KD (~IC50)", "полумаксимальная эффективная концентрация (EC50)", или "полумаксимальная ингибиторная концентрация (IC50)". Измерение приписывалось группе Søren Buus (University of Copenhagen, Дания), если строка "Buus" появлялась в поле "Авторы". В том случае, если поле авторов включало строки "Sette" или "Sidney", измерение приписывалось группе Alessandro Sette (La Jolla Institute for Immunology, США). Все другие измерения обозначались как "прочие". Для нумерации пептидов с сильным связыванием, подсчитывали только пептиды с измеренной аффинностью выше 50 нМ (фиг. 12A). Фиг. 12E включает дополнительные данные с tools.iedb.org/main/datasets/, и приведены пептиды с сильным связыванием с аффинностью <100 нМ.
Конструирование конструкции ДНК
[0657] Последовательности генов для аллелей HLA класса I и HLA класса II идентифицировали посредством веб-страницы IPD-IMGT/HLA (ebi.ac.uk/ipd/imgt/hla) и использовали для конструирования рекомбинантных экспрессирующих конструкций. Для HLA класса I α-цепь подвергали слиянию с C-концевым линкером GSGGSGGSAGG (SEQ ID NO: 10), а затем последовательностью биотин-акцепторной пептидной (BAP) метки GLNDIFEAQKIEWHE (SEQ ID NO: 11), стоп-кодоном и варьирующим штрихкодом ДНК, и клонировали в вектор pSF Lenti (Oxford Genetics, Оксфорд, Великобритания) через участки рестрикции NcoI и XbaI. Конструкции HLA класса II аналогичным образом клонировали в pSF Lenti через участки рестрикции NcoI и XbaI, и они состояли из последовательности β-цепи, слитой на C-конце с последовательностью линкер-BAP из конструкции класса I (SGGSGGSAGGGLNDIFEAQKIEWHE (SEQ ID NO: 12)), за которой следует другой короткий линкер GSG и последовательность рибосомального пропускания F2A (VKQTLNFDLLKLAGDVESNPGP (SEQ ID NO: 13)), последовательность α-цепи, HA-метка (GSYPYDVPDYA (SEQ ID NO: 14)), стоп-кодон и варьирующий штрихкод ДНК. Идентичность всех последовательностей ДНК подтверждали посредством секвенирования по методу Сэнгера.
Клеточная культура и временная трансфекция
[0658] Клетки Expi293 (Thermo Scientific) выращивали в среде Expi293 (Thermo Scientific) c 8% CO2 при 37°C при встряхивании при 125 об/мин. Клетки Expi293 поддерживали при плотности клеток от 0,5×106/мл до 6×106/мл с регулярным пассированием раз в две недели. 30 мл суспензии клеток Expi293 использовали для временной трансфекции при плотности клеток приблизительно 3×106/мл и жизнеспособности >90%. В кратком изложении, 30 мкг ДНК (1 мкг/мл ДНК на мл клеточной суспензии) разбавляли в 1,5 мл среды Opti-MEM (Thermo Scientific) в одной пробирке, в то время как 80 мкл реагента для трансфекции ExpiFectamineTM 293 (Thermo Scientific) разбавляли во второй пробирке, содержавшей 1,5 мл Opti-MEM. Эти две пробирки инкубировали при комнатной температуре в течение пяти минут, объединяли, осторожно перемешивали и инкубировали при комнатной температуре в течение 30 минут. К клеткам Expi293 добавляли смесь ДНК и ExpiFectamine и инкубировали при 37°C, 8% CO2, относительной влажности 80%. Через 48 ч трансфицированные клетки собирали посредством четырех технических повторений в количестве 50×106 клеток на пробирку, центрифугировали, промывали один раз 1×Gibco DPBS (Thermo Scientific) и быстро замораживали в жидком азоте для масс-спектроскопического анализа. Аликвоту 1×106 клеток собирали из каждой подвергнутой трансфекции партии и анализировали посредством вестер-блоттинга с антителом против BAP (Rockland Immunochemicals Inc., Limerick, PA) или антителом против HA (Bio-Rad, Hercules, CA) для подтверждения экспрессии меченного аффинной меткой белка HLA.
[0659] Клетки A375 (ATCC) выращивали в DMEM с 10% FBS и поддерживали в культурах до смыкания монослоя не более чем на 80% с регулярным пассированием. Для экспериментов с использованием масс-спектрометрии клетки A375 культивировали в 500-см2 чашке при плотности посева 18,5×106 клеток/мл в 100 мл, как вычисляют из количества клеток смыкающегося на 70% монослоя. Через 24 часа клетки трансфицировали TransIT-X2 (Mirus Bio, Madison, WI), следуя системному протоколу TransIT с поправкой на общий объем культуры. Через 48 ч клеточную среду аспирировали и клетки промывали 1X Gibco DPBS (Thermo Scientific). Для сбора клетки A375 инкубировали в течение 10 минут при 37°C с 30 мл раствора для неферментативной диссоциации клеток (Sigma-Aldrich), центрифугировали, промывали 1X DPBS и распределяли аликвотами в количестве 50×106 клеток на образец. Клетки 293T и HeLa приобретали от ATCC и культивировали при 37°C при 5% CO2 в DMEM, 10% FBS, 2 мМ L-глутамине или DMEM+10% FBS, соответственно. Оба клеточных линии трансфицировали конструкциями HLA с использованием реагента TransIT LT1 (Mirus Bio, Madison, WI) в соответствии с инструкциями изготовителя и обрабатывали через 48 ч после трансфекции, как описано для клеток A375. Из всех образцов аликвоту 1×106 клеток собирали после каждой трансфекции и анализировали посредством вестерн-блоттинга с использованием антитела против BAP (Rockland Immunochemicals Inc., Limerick, PA) или антитела против HA (Bio-Rad, Hercules, CA) для подтверждения экспрессии меченного аффинной меткой белка HLA.
Экспрессия и очистка белка BirA
[0660] Вектор pET19, кодирующий BirA E. coli, слитый с C-концевой гексагистидиновой меткой (SEQ ID NO: 15). Химически компетентные клетки E. coli BL21 (DE3) (New England Biolabs) трансформировали экспрессирующей BirA плазмидой, выращивали при 37°C в среде LB плюс 100 мкг/мл ампициллина до OD600 0,6-0,8 и охлаждали до 30°C перед индукцией экспрессии добавлением 0,4 мМ изопропил-β-D-тиогалактопиранозида. Выращивание клеток E. coli продолжали при 30°C в течение 4 ч. Клетки E. coli собирали центрифугированием при 8000×g в течение 30 минут при 4°C и хранили при -80°C до применения. Замороженные клеточные осадки, экспрессирующие рекомбинантный BirA, ресуспендировали в буфере IMAC (50 мМ NaH2PO4 pH 8,0, 300 мМ NaCl) с 5 мМ имидазолом, инкубировали с 1 мг/мл лизоцима в течение 20 минут на льду и лизировали посредством обработки ультразвуком. Клеточный дебрис и нерастворимые материалы удаляли центрифугированием при 16000×g в течение 30 минут при 4°C. Затем очищенный супернатант загружали на 5-мл колонку HisTrap HP с использованием хроматографической системы ÄKTA pure (GE Healthcare), промывали буфером IMAC плюс 25 мМ и 50 мМ имидазол, а затем элюировали 500 мМ имидазолом. Фракции, содержавшие BirA, объединяли и подвергали диализу против 20 мМ Tris-HCl pH 8,0 с 25 мМ NaCl и загружали в 5-мл колонку HiTrap Q HP (GE Healthcare) и элюировали посредством применения линейного градиента от 25 до 600 мМ NaCl. Фракции, содержавшие в высокой степени чистый BirA, объединяли, подвергали замене буфера в буфере для хранения (20 мМ Tris-HCl pH 8,0 100 мМ NaCl, 5% глицерин) и концентрировали приблизительно до 5-10 мг/мл, распределяли аликвотами и быстро замораживали в жидком азоте для хранения при -80°C. Концентрацию белка BirA определяли посредством УФ-спектроскопии при OD280 нм с использованием вычисленного коэффициента экстинкции ε=47,440 M-1 см-1.
Протокол вестерн-блоттинга
[0661] Образцы добавляли в буфер для образца XT Sample Buffer и восстановитель XT Reducing Agent (Bio-Rad, Hercules, CA), нагревали при 95°С в течение пяти минут, затем объем, соответствующий ~100000 клеток, наносили на 10% гели Criterion XT Bis-Tris gels (Bio-Rad) и подвергали электрофорезу при 200 В в течение 35 минут с использованием PowerPac Basic Power Supply (Bio-Rad, Hercules, CA) с подвижным буфером XT MES Running Buffer (Bio-Rad, Hercules, CA). Гели кратковременно ополаскивали водой, затем белки переносили на PVDF-мембраны в Invitrogen iBlot Transfer Stacks (Thermo Fisher Scientific) с использованием установки P3 на устройстве Invitrogen iBlot2 Gel Transfer Device (Thermo Scientific). Для мониторинга молекулярной массы использовали Precision Plus Protein All Blue Standard (Bio-Rad, Hercules, CA). Далее мембраны промывали 3 × пять минут посредством буфера Pierce TBS Tween 20 (TBST) (25 мМ Tris, 0,15 мМ NaCl, 0,05% (об./об.) Tween 20, pH 7,5), блокировали в течение 1 ч при комнатной температуре в TBST-M (TBST, содержащая 5% (масс./об.) обезжиренное сухое молоко), затем инкубировали в течение ночи при 4°C в TBST-B (TBST, содержащая 5% (масс./об.) бычий сывороточный альбумин (Sigma Aldrich)] и разведение 1:5000 как антитела кролика против бета-тубулина (каталожный номер # ab6046, Abcam), так и антитела кролика против эпитопной метки биотинлигазы (каталожный номер №100-401-B21, Rockland Immunochemicals). Затем мембраны промывали 3× пять минут посредством TBST, инкубировали в течение 1 ч при комнатной температуре в TBST-M, содержавшей разведение 1:10000 антитела козы против IgG кролика (конъюгированное антитело H+L-пероксидаза хрена (каталожный номер №170-6515, Bio-Rad, Hercules, CA), затем промывали при комнатной температуре 3 × пять раз посредством TBST. Наконец, мембраны инкубировали с субстратом Pierce ECL Western Blotting Substrate (Thermo Fisher Scientific, Rockford, IL), проявляли с использованием устройства для визуализации ChemiDoc XRS+ Imager (Bio-Rad) и визуализировали с использованием программного обеспечения Image Lab (Bio-Rad).
Выделение меченных аффинной меткой комплексов HLA-пептид
[0662] Выделение меченного аффинной меткой комплекса HLA-пептид проводили из клеток, экспрессирующих меченные BAP аллели HLA и отрицательные контрольные клеточные линии, которые экспрессировали только эндогенные комплексы HLA-пептид без BAP-меток. Агарозную смолу в виде гранул с нейтравидином промывали три раза 1 мл холодного PBS, а затем применяли в аффинной очистке HLA-пептида. Замороженные гранулы, содержавшие 50×106 клеток, экспрессировавших BAP-меченные пептиды HLA, размораживали на льду в течение 20 минут и осторожно лизировали посредством пипетирования вручную в 1,2 мл холодного лизирующего буфера [20 мМ Tris-Cl pH 8, 100 мМ NaCl, 6 мМ MgCl2, 1,5% (об./об.) Triton X-100, 60 мМ октилглюкозид, 0,2 мМ 2-йодацетамид, 1 мМ EDTA pH 8, 1 мМ PMSF, 1X свободный от EDTA полный коктейль ингибиторов протеаз (Roche, Basel, Швейцария)]. Лизаты инкубировали с переворачиванием при 4°C в течение 15 минут с >250 единиц нуклеазой Benzonase (Sigma-Aldrich) для деградации ДНК/РНК и центрифугировали при 15000×g при 4°C в течение 20 минут для удаления клеточного дебриса и нерастворимых материалов. Осветленные супернатанты переносили в новые пробирки и BAP-меченные пептиды HLA биотинилировали посредством инкубации с переворачиванием при комнатной температуре в течение 10 минут в 1,5-мл пробирке с 0,56 мкМ биотином, 1 мМ ATP и 3 мкМ BirA. Супернатанты инкубировали с переворачиванием при 4°C в течение 30 минут с объемом, соответствующим 200 мкл взвеси высокоемкой агарозной смолы в виде гранул с нейтравидином Pierce (Thermo Scientific) для аффинного обогащения биотинилированных комплексов HLA-пептид. Наконец, связанную с HLA смолу промывали четыре раза с 1 мл холодного буфера для промывания (20 мМ Tris-Cl pH 8, 100 мМ NaCl, 60 мМ октилглюкозид, 0,2 мМ 2-йодацетамид, 1 мМ EDTA pH 8), затем промывали четыре раза 1 мл холодного 10 мМ Tris-Cl pH 8. Между промываниями связанную с HLA смолу осторожно перемешивали вручную, а затем осаждали центрифугированием при 1500×g при 4°C в течение одной минуты. Промытую связанную с HLA смолу хранили при -80°C или сразу подвергали элюированию HLA-пептидов и обессоливанию.
Выделение комплексов HLA-пептид на основе антител
[0663] Комплексы HLA класса II DR-пептид выделяли из мононуклеарных клеток периферической крови (PBMC) здорового донора. Объем, соответствующий 75 мкл смолы GammaBind Plus Sepharose resin, промывали три раза 1 мл холодного PBS, инкубировали при переворачивании с 10 мкг антитела при 4°C в течение ночи, затем промывали три раза 1 мл холодного PBS, а затем использовали в иммунопреципитации HLA-пептид. Замороженные осадки PBMC, содержавшие 50×106 клеток, размораживали на льду в течение 20 минут и осторожно лизировали посредством пипетирования в 1,2 мл холодного лизирующего буфера [20 мМ Tris-Cl pH 8, 100 мМ NaCl, 6 мМ MgCl2, 1,5% (об./об.) Triton X-100, 60 мМ октилглюкозид, 0,2 мМ 2-йодацетамид, 1 мМ EDTA pH 8, 1 мМ PMSF, 1X полный свободный от EDTA коктейль ингибиторов протеаз (Roche, Basel, Швейцария)]. Лизаты инкубировали с переворачиванием при 4°C в течение 15 минут с >250 единиц нуклеазы Benzonase (Sigma-Aldrich) для деградации ДНК/РНК и центрифугировали при 15000×g при 4°C в течение 20 минут для удаления клеточного дебриса и нерастворимых материалов. Затем супернатанты инкубировали с переворачиванием при 4°C в течение 3 часов с антителом против HLA DR (TAL 1B5, продукт № sc-53319; Santa Cruz Biotechnology, Dallas, TX), связанным со смолой GammaBind Plus Sepharose resin (GE Life Sciences) для иммунопреципитации комплексов HLA DR-пептид. Наконец, смолу со связанным HLA промывали четыре раза с 1 мл холодного буфера для промывания (20 мМ Tris-Cl pH 8, 100 мМ NaCl, 60 мМ октилглюкозид, 0,2 мМ 2-йодацетамид, 1 мМ EDTA pH 8), затем промывали четыре раза 1 мл холодного 10 мМ Tris-Cl, pH 8. Между промываниями смолу со связанным HLA осторожно перемешивали, а затем осаждали центрифугированием при 1500×g при 4°C в течение 1 минуты. Промытую смолу со связанным HLA хранили при -80°C или сразу подвергали элюированию HLA-пептида и обессоливанию.
Элюирование HLA-пептидов и обессоливание
[0664] HLA-пептиды элюировали из меченных аффинной меткой и эндогенных комплексов HLA и одновременно обессоливали с использованием твердофазной системы экстракции Sep-Pak (Waters, Milford, MA). В кратком изложении, кассеты tC18 Sep-Pak Vac 1 cc (50 мг) с размером частиц 37-55 мкм подсоединяли к 24-позиционному распределителю для экстракции (Restek,), активировали два раза 200 мкл MeOH, а затем 100 мкл 50% (об./об.) ACN/1% (об./об.) FA, затем промывали четыре раза 500 мкл 1% (об./об.) FA. Для диссоциации HLA-пептидов из меченных аффинной меткой пептидов HLA и облегчения связывания пептидов с твердой фазой tC18, 400 мкл 3% (об./об.) ACN/5% (об./об.) FA добавляли в пробирки, содержавшие агарозную смолу в виде гранул со связанным HLA. Взвесь перемешивали пипетированием, а затем переносили в кассеты Sep-Pak. Пробирки и наконечники пипеток ополаскивали 1% (об./об.) FA (2×200 мкл) и промывные воды переносили с кассеты. 100 фмоль смеси для калибровки времени удержания Peptide Retention Time Calibration (PRTC) mixture Pierce (Thermo Scientific) добавляли в кассеты в качестве контроля загрузки. Агарозную смолу в виде гранул инкубировали два раза в течение пяти минут с 200 мкл 10% (об./об.) AcOH для дальнейшей диссоциации HLA-пептидов из меченных аффинной меткой HLA-пептидов, а затем промывали четыре раза 500 мкл 1% (об./об.) FA. HLA-пептиды элюировали с tC18 в новые 1,5-мл микропробрки (Sarstedt,) посредством ступенчатого фракционирования с использованием 250 мкл 15% (об./об.) ACN/1% (об./об.) FA, а затем 2×250 мкл 30% (об./об.) ACN/1% (об./об.) FA. Растворы, использованные для активации, загрузки образца, промывания и элюирования, проходили посредством гравитации, однако для удаления оставшегося элюата из кассет использовали вакуум (< -2,5 PSI). Элюаты, содержавшие HLA-пептиды, замораживали, сушили посредством центрифугирования в вакууме, и хранили при -80°С, а затем подвергали второму процессу обессоливания.
[0665] Вторичное обессоливание образцов HLA-пептид проводили с использованием StageTips собственной конструкции, упакованных с использованием двух пуансонов калибра 16 дисков для твердофазной экстракции Empore C18 (3M, St. Paul, MN), как описано ранее. StageTips активировали два раза посредством 100 мкл MeOH, а затем 50 мкл 50% (об./об.) ACN/0,1% (об./об.) FA, а затем промывали три раза посредством 100 мкл 1% (об./об.) FA. Высушенные HLA-пептиды солюбилизировали посредством добавления 200 мкл 3% (об./об.) ACN/5% (об./об.), а затем загружали на StageTips. Пробирки и наконечники пипеток ополаскивали 1% (об./об.) FA (2×100 мкл) и объем промывочной жидкости переносили на StageTips, а затем StageTips промывали пять раз посредством 100 мкл 1% (об./об.) FA. Пептиды элюировали с использованием ступенчатого градиента объемом 20 мкл 15% (об./об.) ACN/0,1% (об./об.) FA, а затем двумя 20-мкл порциями 30% (об./об.) ACN/0,1% (об./об.) FA. Загрузку образцов, промывание и элюирование проводили на настольной центрифуге при максимальной скорости 1500-3000×g. Элюаты замораживали, сушили посредством центрифугирования в вакууме и хранили при -80°С.
Секвенирование HLA-пептидов посредством тандемной масс-спектрометрии
[0666] Во всех анализах nanoLC-ESI-MS/MS использовались те же условия разделения LC, которые описаны ниже. Образцы подвергали хроматографическому разделению с использованием Proxeon Easy NanoLC 1200 (Thermo Scientific, San Jose, CA), оборудованного капиллярами с внутренним диаметром 75 мкм PicoFrit (New Objective, Inc., Woburn, MA) с 10-мкм эмиттером, упакованными под давлением 1000 фунт./кв. дюйм посредством He до ~30-40 см гранулами C18 Reprosil с размером частиц 1,9 мкм /размером пор 200 Å и нагревали при 60°C в ходе разделения. Колонку уравновешивали посредством 10X объемом слоя буфера A (0,1% (об./об.) FA и 3% (об./об.) ACN), образцы загружали в 4-мкл 3% (об./об.) ACN/5% (об./об.) FA, и пептиды элюировали посредством линейного градиента 7-30% буфера B (0,1% (об./об.) FA и 80% (об./об.) ACN) в течение 82 минут, 30-90% буфера B в течение шести минут, а затем удерживали при 90% буфере B в течение 15 минут для промывания колонки. Подгруппу образцов элюировали посредством линейного градиента 6-40% буфера B в течение 84 минут, 40-60% буфера B в течение девяти минут, а затем удерживали при 90% буфера B в течение пяти минут и 50% буфера B в течение девяти минут для промывания колонки. Линейные градиенты для элюирования образцов прогоняли при скорости 200 нл/мин и получали срединную ширину пиков ~13 с.
[0667] В ходе зависимого от данных исследования элюированные пептиды загружали в масс-спектрометр Orbitrap Fusion Lumos (Thermo Scientific), оборудованный источником ионов Nanospray Flex Ion source (Thermo Scientific) при 2,2-2,5 кВ. Полное сканирование MS проводили при разрешении 60000 с m/z от 300 до 1700 (AGC Target 4e5, максимальный IT 50 мс). После каждого полного сканирования следовало время цикла 2 с или наилучшие 10 зависимых от данных сканирований MS2 при разрешении 15000, с использованием ширины выделения 1,0 m/z, энергии столкновений 34 (данные для HLA класса) и 38 (данные для HLA класса II), ACG Target 5e4 и максимального времени заполнения 250 мс. Ширину выделения 1,0 m/z использовали, поскольку пептиды HLA класса II имеют тенденцию к большей длине (срединное значение 16 аминокислот с подгруппой пептидов из >40 аминокислот), так что моноизотопный пик не всегда является наиболее высоким в кластере изотопов, и программное обеспечение для получения данных масс-спектрометра помещает наиболее высокий изотопный пик в центр окна выделения в отсутствии указанного смещения. Таким образом, окно выделения 1,0 m/z позволяет совместное выделение моноизотопного пика, даже когда он не является наиболее высоким пиком в изотопном кластере, поскольку зарядовые состояния пептидов класса II часто представляют собой +2 или выше. Было разрешено динамическое исключение с количеством повторов 1 и длительностью исключения 5 с, чтобы позволить ~3 PSM на выбранного предшественника. Изотопы исключали, в то время как зависимые сканирования на однозарядовое состояние на предшественника было выключено, поскольку идентификация HLA-пептидов основана на качестве PSM, так что множество PSM для различных зарядовых состояний далее повышают уверенность авторов настоящего изобретения в идентификации пептидов. Скрининг зарядовых состояний для получения данных для HLA класса II был разрешен вместе с моноизотопным отбором предшественников (MIPS) с использованием режима Peptide Mode для предотвращения запуска MS/MS на ионах предшественников с зарядовым состоянием 1 (только для аллелей с основными якорными остатками), >7 или без присвоения. Для сбора данных HLA класса I, отбирали ионы предшественников с зарядовым состоянием 1 (диапазон масс 800-1700 m/z) и 2-4, в то время как зарядовые состояния >4 и без присвоения были исключены.
[0668] Детекцию пептидов с использованием спектрометрии подвижности ионов в несимметричном поле высокой напряженности (FAIMS) оценивали с использованием следующего протокола. Эндогенно процессированные и презентированные пептиды HLA класса I и HLA класса II из клеток A375 подвергали как кислотному обращенно-фазовому (aRP), так и основному обращенно-фазовому (bRP) фракционированию вне системы перед анализом nLC-MS/MS с использованием масс-спектрометра Orbitrap Fusion Lumos Tribid с интерфейсом FAIMS или без него. Процесс продемонстрирован на фиг. 42A. На фиг. 42B, представлено сопоставление FAIMS с низкими количествами трипсинизированных образов клеток Jurkat, клеток HeLa. Пептиды, связывающие как HLA класса I, так и HLA класса II, анализировали с использованием FAIMS (фиг. 43A и фиг. 43B; и фиг. 44A и фиг. 44B соответственно). В каждом случае происходило небольшое усовершенствование детекции пептидов, особенно в случае пептидов, фракционированных посредством bRP. На фиг. 45 и фиг. 46 представлен размер пересечения.
Интерпретация данных LC-MS/MS
[0669] Этот раздел касается, например, фиг. 29. Спектры масс интерпретировали с использованием пакета программ Spectrum Mill v6.0 pre-Release (Agilent Technologies, Santa Clara, CA). Спектры MS/MS исключали из поиска, если они не имели MH+ предшественника в диапазоне 600-2000 (класс I)/600-4000 (класс II), имели заряд предшественника > 5 (класс I)/ >7 (класс II) или имели минимум <5 обнаруженных пиков. Объединение сходных спектров с одним и тем же m/z предшественника, полученным в одном и том же хроматографическом пике, было выключено. Для спектров MS/MS проводили поиск против базы данных, которая содержала гены UCSC Genome Browser с аннотацией hg19 генома и их кодирующие белок транскрипты (63691 записей; 10917867 уникальных 9-мерных пептидов) в комбинации с 264 часто встречающимися примесями. Перед поиском в базе данных все MS/MS должны были проходить спектральный фильтр качества длиной последовательности-метки >2, например, минимум из 3 масс, разделенных массой аминокислоты внутри цепи. Было установлен минимальный показатель расщепления основной цепи (BCS) 5, и использовалась оценочная схема ESI QExactive HLAv2. Для всех спектров нативных образцов HLA-пептидов, не восстановленных и алкилированных, проводили поиск с использованием отсутствия ферментной специфичности, фиксированной модификацией цистеина в качестве цистеинилирования, со следующими переменными модификациями: окисленный метионин (m), пироглутаминовая кислота (N-концевая q), карбамидометилирование (c). Для восстановленных и алкилированных образцов HLA-пептид проводили поиск с использованием отсутствия ферментной специфичности, фиксированной модификации цистеина в качестве карбамидометилирования со следующими переменными модификациями: окисленный метионин (m), пироглутаминовая кислота (N-концевой q), цистеинилирование (c). Как для нативных, так и для восстановленных и алкилированных наборов данных HLA-пептид использовали допуск по массе предшественника ±10 м.д., допуск по массе продукта ±10 м.д., и минимальную интенсивность соответствующего пика 30%. Соответствие пептидных спектров (PSM) для индивидуальных спектров автоматически обозначалось как достоверно присвоенное с использованием модуля аутовалидации Spectrum Mill для применения оценки FDR на основе мишени-ловушки в ранге PSM для установки оценочных пороговых критериев. Стратегия автоматических пороговых значений с использованием минимальной длины последовательности 7, автоматической фильтрацией массы предшественников вариабельного диапазона, и оценки и пороговых значений оценки дельта ранг 1 - ранг 2, оптимизированных для всех прогонов LC-MS/MS для аллеля HLA, обеспечивала оценку FDR PSM <1,0% для каждого зарядового состояния предшественника.
[0670] Идентифицированные пептиды, которые проходили оценку FDR PSM <1,0%, далее фильтровали в отношении примесей путем удаления всех пептидов, отнесенных к 264 распространенным белкам-примесям в эталонной базе данных, и путем удаления пептидов, идентифицированных в аффинном анализе соосаждения MAPTAC™ для отрицательного контроля. Кроме того, все идентифицированные пептиды, которые картировались на трипсинизированном продукте расщепления in silico эталонной базы данных, удаляли, поскольку эти пептиды не могут быть исключены из триптических примесей в результате переноса образца на колонку uPLC.
Моноаллельное распределение гетеродимеров HLA-DR, -DQ, DP с использованием протокола MAPTAC™
[0671] Моноаллельное отнесение HLA к идентифицированным посредством LC-MS/MS пептидам проводили посредством двух подходов. Поскольку аллельное варьирование HLA-DRA1 ограничено и считается, что оно не влияет на связывание пептидов, все данные из экспериментов с DR (определение профиля DRB1, 3, 4 и 5) считались моноаллельными, что означало, что пептиды, наиболее вероятно, связывались с гетеродимерами HLA класса II, содержащими улавливающие бета-цепи в паре с улавливающими альфа-цепями. Однако остается возможность, что некоторые пептиды могут связываться с гетеродимерами HLA II, содержащими нокинные бета-цепи в паре с определенными эндогенно экспрессируемыми альфа-цепями.
[0672] Напротив, для локусов HLA-DP и HLA-DQ, альфа-цепи демонстрируют значимые аллельные варианты, так что присутствие как нокинных, так и эндогенных аллелей альфа-цепей, создает потенциал для множества гетеродимеров. Например, нокинные альфа- и бета-цепи, кодирующие определенные гетеродимеры HLA-DP и HLA-DQ, в каждом случае могут образовывать пару с эндогенно экспрессируемыми альфа- и бета-цепями, образующими четыре уникальных гетеродимера для каждой конструкции HLA-DP и HLA-DQ MAPTAC™. Таким образом, специфичность связывания среди очищенных популяций пептидов MAPTAC™ не является моноаллельной. Для уменьшения этой проблемы эндогенного образования пар использовали конструкцию, лишенную альфа-цепи, которая (при отсутствии нокинной альфа-цепи) позволила авторам изобретения идентифицировать популяцию пептидов, которые вероятно связываются с гетеродимерами HLA, содержащими эндогенные альфа-цепи и бета-цепи MAPTAC™. Эти пептиды вычислительно вычитали из соответствующих экспериментов MAPTAC™ с альфа + бета-цепями для аппроксимации к популяции пептидов, специфичных к моноаллельной комбинации альфа + бета MAPTAC™.
[0673] Каждый пептид относили к одному или более кодирующим белок транскриптам в аннотации гена UCSC hg19. Поскольку множество идентификаций пептидов перекрываются с другими и, таким образом, составляют по большей части избыточную информацию, пептиды распределяли на "вложенные множества", причем подразумевалось, что каждое из них соответствует ~1 уникальному событию связывания, как показано на фиг. 11C. Например, все из пептидов GKAPILIATDVASRGLDV (SEQ ID NO: 16), GKAPILIATDVASRGLD (SEQ ID NO: 17) и KAPILIATDVASRGLDV (SEQ ID NO: 18) содержат консервативную последовательность KAPILIATDVASRGLD (SEQ ID NO: 19), и, возможно, все из них связывают MHC на том же уровне. Для вложения пептидов данного набора данных строили график, в котором каждый узел соответствовал уникальному пептиду, и создавали границу между любой парой пептидов, имеющих по меньшей мере один 9-мер и картируемых по меньшей мере на одном общем транскрипте. Команду "clusters" в пакете R igraph использовали для идентификации кластеров связанных узлов, и каждый кластер был идентифицирован как вложенное множество. Эта методика гарантирует, что любые два пептида, которые удовлетворяют пограничному критерию (≥1 обычного 9-мера и ≥1 обычного транскрипта) распределяются в одно то же вложенное множество.
Анализ ранее опубликованных данных MS
[0674] Следующий раздел относится по меньшей мере к фиг. 12A-12F, фиг. 35, фиг. 36A-36B, фиг. 38D, фиг. 39A-39C, фиг. 40A-40B. Опубликованные наборы данных LC-MS/MS, которые предоставляли файлы.raw, подвергали повторной обработке с использованием пакета программ Spectrum Mill v6.0 pre-Release (Agilent Technologies, Santa Clara, CA). Наборы данных, которые были получены на устройстве Thermo Orbitrap (например, Velos, QExactive, Fusion, Lumos), в которых использовалась фрагментация HCD и набор данных MS и MS/MS в Orbitrap (высокое разрешение), анализировали с использованием параметров, описанных в приведенном выше разделе "Интерпретация данных LC-MS/MS". Для наборов данных MS и MS/MS высокого разрешения, в которых использовалась фрагментация CID, те же параметры, которые описаны выше, использовались со схемой оценки ESI Orbitrap. Для наборов данных с коллекцией данных MS в орбитрэпе и коллекцией данных MS/MS в ионной ловушке, следующие те же самые параметры, которые описаны выше, также использовались со следующими отклонениями. Для данных HCD использовали схему оценки ESI QExactive HLAv2, в то время как для данных CID использовали схему оценки ESI Orbitrap. Использовали допуск по массе предшественника ±10 м.д., допуск по массе продукта ±0,5 Да. Для наборов данных MS/MS как высокого разрешения, так и низкого разрешения, соответствие пептидных спектров (PSM) для индивидуальных спектров автоматически обозначалось как достоверно присвоенное с использованием модуля аутовалидации Spectrum Mill для применения оценки FDR на основе мишень-ловушка в ранге PSM для установки оценочных пороговых критериев. Стратегия автоматических пороговых значений с использованием минимальной длины последовательности 7, автоматической фильтрацией массы предшественников вариабельного диапазона, и оценки и пороговых значений оценки дельта ранг 1 - ранг 2, оптимизированные для всех прогонов LC-MS/MS для аллеля HLA, обеспечивала оценку FDR PSM <1,0% для каждого зарядового состояния предшественника.
[0675] Частоты аминокислот в протеоме человека вычисляли на основе последовательностей для всех кодирующих белок генов в аннотации UCSC hg19 (выбор одного транскрипта случайным образом для генов, соответствующих множеству изоформам транскриптов), как показано на фиг. 11D. Частоты IEDB определяли посредством идентификации уникального набора пептидов по меньшей мере с одним наблюдением аффинности ≤50 нМ (исключая некоторые пептиды c гексавалентным полигистидином на их C-конце). Частоты MAPTAC™ сначала рассматривали в контексте стандартного протокола прямой фазы для пяти аллелей DRB1 (DRB1*01:01, DRB1*03:01, DRB1*09:01, и DRB1*11:01), с использованием только одного пептида (наиболее длинного) на вложенное множество. Кроме того, частоты MAPTAC™ отдельно вычисляли для основного протокола обратной фазы среди нескольких аллелей. Данные MS из внешних наборов данных анализировали независимо от потенциального источника аллеля и аналогично с использованием наиболее длинного пептида на вложенное множество.
Конструирование логотипов последовательностей для класса I (пептид, связывающий HLA класса I)
[0676] Для каждого аллеля класса I (как представлено на фиг. 14A), создавали логотип последовательности длиной 9 посредством определения профилей аминокислотных частот в первых пяти положениях (картирование положений логотипов 1-5) и последних четырех положениях (картирование положения логотипов 6-9) соответствующих пептидов. Таким образом, пептиды вносили вклад в логотипы последовательностей независимо от их длины. Как и в случае логотипов для класса II, высота букв пропорциональна частоте каждой аминокислоты в каждом положении, и для положений с низкой энтропией используется кодирование цветом.
Спрогнозированная аффинность выявленных посредством MS пептидов
[0677] Этот раздел связан по меньшей мере с фиг. 14C. Для каждого аллеля класса II все уникальные пептиды длиной от 14 до 17 идентифицировали и оценивали в отношении потенциала связывания с использованием NetMHCIIpan. Для сравнения проводили отбор случайных совпадающих по длине пептидов из протеома человека. Распределение плотностей (как представлено на фиг. 14C) определяли на основе логарифмически преобразованных величин. Некоторые аллели исключали из анализа, поскольку NetMHCIIpan не поддерживает прогнозирование для них.
Измеренная аффинность для выявленных посредством MS пептидов
[0678] Пептиды отбирали для измерения аффинности, если они имели плохую спрогнозированную аффинность связывания NetMHCIIpan (>100 нМ для DRB1*01:01 или >500 нМ для DRB1*09:01 и DRB1*11:01) или если они имели ≤2 эвристически определенных якорей для тестирования в ранее опубликованном биохимическом анализе аффинности MHC-пептид.
Установление обучающих, настраивающих и тестовых частей протеома
[0679] Этот раздел связан по меньшей мере с фиг. 15A (также см. фиг. 31A). Строили график, в котором каждый узел соответствует кодирующему белок транскрипту и присутствуют границы между всеми парами транскриптов, обладающими по меньшей мере 5 уникальными 9-мерми аминокислотного содержимого последовательности (аннотация генов UCSC hg19). Команду "clusters" в пакете R igraph (cran.r-project.org/web/packages/igraph/citation.html) использовали для идентификации кластеров связанных узлов, и каждый кластер был идентифицирован как "группа транскриптов". Таким образом, если два транскрипта имели общую границу (≥5-общих 9-меров), они гарантированно распределялись в одну и ту же группу транскриптов. Из группы транскриптов проводили случайный отбор, помещая 75% в обучающую часть, 12,5% в часть настройки, и 12,5% в тестовую часть. Во всех анализах выявленные посредством MS пептиды (и не выявленные пептиды-ловушки) помещали в части (обучение, настройка или тест) в соответствии с частью их исходных транскриптов. В очень редких случаях, в которых исходные транскрипты картировались на множестве различных положений, предпочтительный порядок распределения представлял собой обучение (наиболее предпочтительный), настройку, а затем тест (наименее предпочтительный). То же распределение протеома использовали во всех способах анализа на основе распределения. Графический подход к распределению протеома использовали для минимизации вероятности того, что сходные пептидные последовательности появятся в ходе обучения и оценки, что может искусственно увеличить эффективность прогнозирования.
Архитектура и обучение средства прогноза связывания на основе CNN для класса II
[0680] Что касается по меньшей мере фиг. 15A, в то время как аминокислоты могут быть представлены посредством "прямого унитарного" кодирования, другие предпочитали кодировать аминокислоты с использованием матрицы PMBEC и матрицы BLOSUM, в которых сходные аминокислоты имеют сходные профили признаков. Для целей фичеризации пептидов, осуществленной авторами изобретения, получали уникальную матрицу на основе близости аминокислот в решенных белковых структурах. Идея этого подхода состоит в том, что типичные соседи аминокислот должны отражать ее химические свойства. Для каждой аминокислоты в каждой из ~100000 белковых структур DSSP (cdn.rcsb.org/etl/kabschSander/ss.txt.gz) определяли остаток, который был наиболее близким в 3D-пространстве, но находился на расстоянии по меньшей мере 10 аминокислот в первичной последовательности. С использованием этих данных определяли количество раз, когда ближайшим соседом аланина был аланин, количество раз, когда ближайшим соседом аланина был цистеин и т.д., с получением матрицы величин близости 20×20. Каждый элемент матрицы делили на результат умножения суммы соответствующих ему столбцов и рядов, и всю матрицу подвергали логарифмическому преобразованию. Наконец, из каждого элемента вычитали среднюю величину для всей матрицы. Три дополнительных физических признака - гидрофобность, заряд и размер - добавляли в качестве дополнительных столбцов, так что каждая аминокислота была представлена 23 входными признаками.
Сопоставление эффективности прогнозирования для выявленных посредством MAPTAC™ пептидов, связанное с фиг. 21A-B
[0681] Для оценки эффективности прогнозирования для данного аллеля было необходимо определить набор пептидов, которые могли быть выявлены (поскольку они присутствуют в протеоме), но не выявлены в данных MS. Эти негативные примеры назвали "естественными ловушками" (в противоположность "перемешанным ловушкам", описанным выше). В качестве основных принципов, было решено следующее:
1. Распределение длины естественных ловушек должно соответствовать распределению длины выявленных посредством MS наиболее подходящих последовательностей.
2. Естественные ловушки не должны содержать последовательность, дублирующую другие естественные ловушки.
3. Естественные ловушки не должны перекрываться с наиболее подходящими последовательностями.
4. Естественные ловушки должны происходить из генов, в которых присутствовала по меньшей мере одна наиболее подходящая последовательность.
[0682] Был создан следующий псевдокод, отражающий процесс оценки, удовлетворяющий этим принципам:
Инициализировать два пустых перечня наиболее подходящих последовательностей, Нминимальный и Нисчерпывающий
Для каждого вложенного множества S выявленных посредством MS пептидов:
Если ни один из пептидов в S не может быть картирован на транскрипте в части обучения или настройки:
Добавить наиболее короткий пептид в S к Нминимальный
Добавить все пептиды в S к Нисчерпывающий
Инициализировать пустой перечень пептидов-ловушек, D
Для каждого кодирующего белок транскрипта (наиболее длинный первый, наиболее короткий последний) в тестовой части:
Если ни один из пептидов в Нисчерпывающий не картируется на транскрипте:
Перейти к следующему транскрипту
Охватить белковую последовательность транскрипта набором перекрывающихся пептидов P, где длина пептидов случайным образом выбрана из распределения длин Нминимальный. Перекрывание составляет 8 аминокислот (Последний пептид в P, как правило, выступает за пределы конца белка).
Пока последний пептид в P все еще выступает:
Вычесть 1 аминокислоту из длины наиболее длинного пептида в P
Для каждого пептида в P:
Если он не имеет 9-мера с пептидом в Нисчерпывающий и он содержит 9-мер, не наблюдаемый ни в одном из пептидов в D:
Добавить пептид к D
В ином случае:
Отбраковать пептид
Нминимальный и D составляют набор данных для оценки
[0683] Для оценки эффективности в этом наборе все n наиболее подходящих пептидов оценивали посредством средства прогнозирования (neonmhc2 или NetMHCIIpan) и оценивали среди набора из 19n ловушек (случайным образом отобранные без замены из полного набора ловушек). Наивысшие 5% пептидов в объединенном наборе называли положительными результатами, и положительную прогностическую величину (PPV) вычисляли в качестве доли положительных результатов, которые представляли собой наиболее подходящие последовательности. Следует отметить, что, поскольку количество положительных результатов ограничивается количеством, равным количеству наиболее подходящих последовательностей, отзыв равен PPV в этом сценарии оценки. Применение постоянного соотношения 1:19 среди аллелей помогает стабилизировать значения эффективности, на которые в ином случае в высокой степени влияет количество наиболее подходящих последовательностей, выявленных для каждого аллеля. Это было сочтено приемлемым, поскольку было предположено, что количество наиболее подходящих последовательность в большей степени связано с экспериментальными условиями и количеством повторений, а не со свойствами, присущими аллелю. Соотношение 1:19 не отклонялось значительно от того, чтобы бы использовалось, если бы уменьшение выборки не осуществлялось.
Сопоставление эффективности прогнозирования при измерении аффинности IEDB
[0684] Что касается фиг. 15B, поскольку NetMHCIIpan обучено на данных аффинности IEDB, его вневыборочную эффективность оценивали с использованием несколько устаревшей версии и показателей IEDB. Соответствие спрогнозированной и измеренной аффинности определяли посредством коэффициента тау Кендалла. Ту же самую статистику использовали для оценки эффективности neonmhc2 на том же наборе показателей. Оценку проводили отдельно по аллелю и по опубликовавшей группе (Sette или Buus).
Сопоставление эффективности прогнозирования естественных CD4+ T-клеточных ответов
[0685] Поскольку значительное большинство ответов CD4+ T-клеток, документированных в IEDB, имеют неизвестную или вычислительно установленную рестрикцию для аллелей касса II, подгруппа записей была сфокусирована на том, что было подтверждено экспериментально посредством тетрамера класса II. Практически все такие записи были депонированы William Kwok Laboratory (Benaroya Research Institute, Seattle, WA), которые используют кровь иммунореактивных индивидуумов для проведения осуществляемого посредством тетрамера картирования эпитопов (TGEM) для различных патогенов и аллергенов. Поскольку негативные пептиды были опубликованы для некоторых исследований, но не для других, изучали исходные публикации для восстановления полного набора положительной и отрицательной реактивности пептидов. Все 20-мерные пептиды оценивали посредством neonmhc2 и NetMHCIIpan. Для вычисления положительной прогностической величины (PPV) среди аллелей сравнимым образом, отрицательные примеры для каждого аллеля случайным образом подвергали понижению выборки до соотношения положительных и отрицательных 1:19. PPV вычисляли в качестве доли экспериментально подтвержденных положительных примеров среди 5% пептидов с наивысшей оценкой. Эффективность также оценивали посредством кривых зависимости чувствительности от частоты ложноположительных заключений.
Оценка эффективности обратной свертки для пептидов MHC II
[0686] Для оценки способности инструмента GibbsCluster (v2.0) к кластеризации мультиаллельных данных для пептидов MHC класса II по аллелю происхождения, сначала рассматривали пептиды из многообразного набора опубликованных DR-специфических экспериментах на индивидуумах с известным генотипом DR (таблица 2). В некоторых случаях в оригинальной публикации было представлено типирование HLA-DRB1, но пропущено типирование для HLA-DRB3/4/5. Для этих случаев было рассматривали сцепление DR1:DR3/4/5, предоставляемое IMGT, и, если этого было недостаточно для четырехзначного типирования, использовали сцепление, наблюдаемое в популяции "USASanFranciscoCaucasian" (allelefrequencies.net, ID популяции 3098: таблица 2).
Таблица 2
идентичен DRB4*01:03
(ebi.ac.uk/cgi-bin/ipd/imgt/hla/get_allele.cgi?DRB4*0101)
[0687] Для каждого аллеля DRB1/3/4/5, присутствующего в каждом (расчетном) генотипе, было внесено двадцать пептидов из моноаллельных данных MAPTAC™, полученных авторами изобретения. Затем эти усиленные наборы данных предоставлялись в GibbsCluster-v2.0.
Охарактеризация наблюдаемых участков расщепления пептидов MHC II
[0688] В настоящем описании описан большой набор данных естественным образом процессируемых и презентируемых пептидов MHC II путем объединения идентификаций пептидов среди нескольких исследований, в которых использовалась иммунная очистка для определения профиля в тканях человека (таблица 2). Поскольку многие пептиды имеют одинаковый N-конец (например, GKAPILIATDVASRGLDV (SEQ ID NO: 16) и GKAPILIATDVASRGLD (SEQ ID NO: 17)) или одинаковый C-конец (например, GKAPILIATDVASRGLD (SEQ ID NO: 18) и KAPILIATDVASRGLD (SEQ ID NO: 19)), исследовали два набора неизбыточных участков расщепления, один для N-концов и один для C-концов. Затем эквивалентное количество уникальных невыявленных N-концевых и C-концевых участков расщепления отбирали случайным образом из набора генов, которые продуцировали по меньшей мере один пептид MHC II. Эти четыре набора данных обозначали как N-концевые наиболее подходящие последовательности, C-концевые наиболее подходящие последовательности, N-концевые ловушки и C-концевые ловушки. Кроме того, для указания на положения выше пептидов, в пептидах и ниже пептидов, использовали систему наименования, которая представлена на фиг. 41.
[0689] Частоту каждой аминокислоты определяли для положений U10-N3 для N-концевых наиболее подходящих последовательностей, и эти частоты сравнивали с частотами, наблюдаемыми для N-концевых ловушек. Для определения того, продемонстрировали ли наиболее подходящие последовательности и ловушки значимое отличие в частоте данной аминокислоты в данном положении, создавали таблицу 2×2 (например, количество наиболее подходящих последовательностей, для которых U1 представляет собой лизин, количество ловушек, для которых U1 представляет собой лизин; количество наиболее подходящих последовательностей, для которых U1 не является лизином, и количество ловушек, для которых U1 не является лизином) и оценивали посредством критерия хи-квадрат. Аналогичный подход использовали для анализа частот аминокислот в положениях C3-D10 C-концевых наиболее подходящих последовательностей и ловушек.
[0690] Второй анализ учитывал статистическое сцепление между остатками, непосредственно предшествующими и следующими после событий расщепления. Во-первых, количество пар U1:N1 (A:A, A:C, A:D, …, Y:V, Y:W, Y:Y) сравнивали для N-концевых наиболее подходящих последовательностей против N-концевых ловушек, и значимость увеличенной/сниженной представленности для каждой пары определяли посредством критерия хи-квадрат для таблицы сопряженности 2×2 (например, количество наиболее подходящих последовательностей с P:K, количество ловушек с P:K; количество наиболее подходящих последовательностей без P:K, количество ловушек без P:K). Аналогичный подход использовали для анализа частот для пар C1:D1 C-концевых наиболее подходящих последовательностей и ловушек.
Сопоставление эффективности различных средств прогнозирования расщепления для класса II
[0691] Периферическую кровь от здоровых доноров подвергали определению профиля для DR-связывающих пептидов. Эти образцы использовали для сопоставления способности связанных с расщеплением переменных/средств прогнозирования для усиления идентификации презентируемых эпитопов класса II.
[0692] Для создания интегрированных средств прогноза, которые прогнозируют презентацию пептидов с использованием как потенциала к связыванию, так и потенциала к расщеплению, сначала конструировали набор данных с использованием того же подхода, который был описан для фиг. 4, но с использованием части "настройки" вместо "тестовой" части. В кратком изложении, это подразумевает использование соотношения наиболее подходящих последовательностей и ловушек 1:20, где ловушки совпадают по длине с наиболее подходящими последовательностями и случайным образом отобраны из набора генов, которые имели по меньшей мере одну наиболее подходящую последовательность. Потенциал к связыванию вычисляли с использованием neonmhc2, и, поскольку эти образцы были мультиаллельными, за показатель связывания для каждого пептида-кандидата принимали среднее значение для 1-4 аллелей DR, определяемых генотипом каждого донора. Этот процесс аппроксимации определял относительный вес, который будет присвоен переменным связывания и расщепления при прямом прогнозировании.
[0693] Для определения эффективности прямого прогнозирования проводили оценку наиболее подходящих последовательностей и ловушек (соотношение 1:19) из "тестовой" части с использованием того же протокола, который был описан непосредственно выше. PPV вычисляли аналогично тому, как и для фиг. 4. Несколько различных средств прогноза расщепления оценивали, как показано в таблице 3.
Таблица 3
Для оценки пептида-кандидата сначала определяли 9-мерную центральную часть. Далее вычисляли максимальный показатель PSM для трех областей: 10 аминокислот выше центральной части (независимо от положения истинного N-концевого участка расщепления), центральная последовательность, и 10 аминокислот после центральной последовательности. Обучение логистической регрессии проводили на части настройки (см. выше) с использованием показателя связывания neonmhc2, а также трех область-специфических показателей PSM.
В пакете программ SCRATCH (Cheng J, Randall AZ, Sweredoski MJ & Baldi P (2005) SCRATCH: a protein structure and structural feature prediction server. Nucleic Acids Res. 33: W72-6, доступно на: dx.doi.org/10.1093/nar/gki396), инструмент ACCpro20 использовали для прогнозирования относительной доступности для растворителя (RSA), и инструмент SSpro8 использовали для прогнозирования признаков вторичной структуры (H: альфа-спираль G: 310-спираль I: пи-спираль E: удлиненная цепь B: бета-мостик T: поворот S: изгиб C: прочее). Аналогично определяли оценку нарушения последовательности на остаток (d2p2.pro/download) по всему протеому, оценивая по шкале 0-5 в соответствии с количеством двигателей прогнозирования, обозначающих положение как нарушенное (использованные серверы: anchor, espritz-d, espritz-n, espritz-x, iupred-l и iupred-s). Наконец, топологические домены (трансмембранный, сигнальный, внеклеточный, просветный и дисульфидный) определяли на уровне остатка с использованием Uniprot (uniprot_sprot.dat). Для каждого пептида-кандидата вычисляли среднее значение RSA и средний показатель нарушения на протяжении 21-мерного окна, центрированного на спрогнозированной 9-мерной центральной части. На протяжении того же окна из 21 остатка вычисляли процентный состав для признаков вторичной структуры и топологических признаков. Четыре модели логистической регрессии были обучены на части "настройки" с использованием следующих входных признаков:
1. Показатель связывания Neonmhc2 и доступность для растворителя (RSA)
2. Показатель связывания Neonmhc2 и оценка нарушения
3. Показатель связывания Neonmhc2 и восемь происходящих из SSpro8 признаков, отражающих вторичную структуру
4. Показатель связывания Neonmhc2 и пять признаков Uniprot, отражающих топологию
Идентификацию пептидов на основе MS объединяли среди множества мультиаллельных экспериментов для класса II из общедоступных источников. Создавали новый признак, отражающий перекрывание пептида-кандидата с одним из ранее выявленных пептидов. В частности, признак принимали за 1, если он имел по меньшей мере один общий 9-мер с любым пептидом в большой выборке из ранее выявленных лигандов MHC II; в ином случае признак принимали за 0. Для логистической регрессии проводили обучение на части "настройки" с использованием показателя связывания neonmhc2 и признака перекрывания.
Взаимосвязь между презентацией посредством MHC класса II и экспрессией
[0694] Пептиды объединяли среди ранее опубликованных экспериментов с использованием MS, которые определяли профиль лигандомов HLA-DR в тканях яичника человека. Для каждого образца с доступными данными RNA-Seq, исходные fastqs загружали из SRA и выравнивали с транскриптомом UCSC hg19 с использованием bowtie2. Количественное определение для генов на уровне транскриптов проводили с использованием количества транскриптов на миллион (TPM), как вычисляли посредством RSEM. Оценки экспрессии далее обрабатывали путем суммирования для генного уровня, исключения некодирующих генов и перенормализации, так чтобы общая сумма TPM достигала 1000000 (перенормализация среди кодирующих белок генов учитывает варьирование от библиотеки к библиотеке уровня nc-РНК).
[0695] Для каждого гена в каждом образце ткани рассматривали его уровень экспрессии в образце и то, продуцировал ли он по меньшей мере один пептид в образце. Среди всех экспериментов с использованием MS, эти наблюдения сортировали в соответствии с уровнем экспрессии и статусом образования пептидов (см. фиг. 18A).
Идентификация генов, представленных на повышенном и сниженном уровне
[0696] Для идентификации генов, представленных на повышенном и сниженном уровне в лигандомах MHC класса II, объединяли данные пяти предшествующих испытаний, которые определяли профиль в ткани яичника, ткани ободочной и прямой кишки и кожной меланомы, рака легкого и рака головы и шеи. Для каждого гена исходным предположением авторов настоящего изобретения было то, что он должен обеспечивать пептиды пропорционально его длине, умноженной на его уровень экспрессии. Для определения длины каждого гена подсчитывали уникальные 9-меры среди всех изоформ транскриптов. Экспрессию на генном уровне получали путем суммирования среди изоформ транскриптов. Наблюдаемое количество пептидов, картированных на каждом гене, определяли на уровне вложенного множества (например, пептиды GKAPILIATDVASRGLDV (SEQ ID NO: 16), GKAPILIATDVASRGLD (SEQ ID NO: 17) и KAPILIATDVASRGLDV (SEQ ID NO: 18) подсчитывались как одно наблюдение).
[0697] Многие образцы из исследования для яичника имели соответствующие данные RNA-Seq, но некоторые не имели. В этих случаях экспрессию оценивали, усредняя среди образцов с доступными данными RNA-Seq. Для исследований рака ободочной и прямой кишки и меланомы отсутствовали соответствующие данные для RNA-Seq для любых образцов, таким образом средние значения вычисляли среди суррогатных образцов с использованием данных из GTEx и TCGA. Во всех случаях исходные fastqs получали и выравнивали, и количественно определяли в соответствии с теми же протоколами, которые описаны выше для RNA-Seq в исследовании для яичника.
[0698] Создавали две матрицы, соответствующие ожидаемым и наблюдаемым количествам, обозначаемые как E и O, соответственно, где строки соответствуют генам и столбцы соответствуют образцам. Сначала заполняли матрицу E путем умножения длины каждого гена на его экспрессию в каждом образце; затем столбцы E подвергали перемасштабированию, чтобы суммы для столбцов E соответствовали суммам для столбцов O. Наконец, проводили анализ на генном уровне путем сравнения сумм строк E и сумм строк O. Гены отмечали в соответствии с их присутствием и концентрацией в плазме человека.
Анализ генов, связанных с аутофагией
[0699] Было идентифицировано два набора генов, связанных с аутофагией. Первый набор включал белки, экспериментально идентифицированные в качестве физических партнеров по взаимодействию для известных связанных с аутофагией генов. Для каждого канонического связанного с аутофагией гена (genenames.org/cgi-bin/genefamilies/set/1022), использованного в качестве приманки в эксперименте IP-MS, депонированного в базе данных Autophagy Interaction Network (доступно на besra.hms.harvard.edu/ipmsmsdbs/cgi-bin/downloads.cgi), были идентифицированы наилучшие 100 идентификаций белков в соответствии со степенью уверенности "WD" (besra.hms.harvard.edu/ipmsmsdbs/cgi-bin/tutorial.cgi). Посредством объединения 22 экспериментов был получен набор из 1004 уникальных генов, достоверно ассоциированных по меньшей мере с одним каноническим связанным с аутофагией геном (фиг. 18C).
[0700] Второй набор связанных с аутофагией генов был идентифицирован с использованием исследования, которое измеряло распространенность белков по всему протеому в эпителиальных клетках детенышей мыши (iBMK) до и после нокаута ATG5 с использованием SILAC (sciencedirect.com/science/article/pii/S1097276514006121). Гены с t-статистикой >5 классифицировали как стабилизированные посредством нокаута ATG5 (условия до голодания; переменная "Intercept_t" в файле дополнительных данных mmc2.xls). Для картирования каждой последовательности мыши в Uniprot ID на hg19 UCSC ID, определяли белковую последовательность человека UCSC, с которой последовательность мыши Uniprot имела наибольшее количество общих 9-меров (фиг. 18C).
[0701] Исходя из фиг. 18B, вычисляли соотношение R для выявленных и ожидаемых пептидов на ген, добавляя псевдоколичество от одного до обоих из числителя и знаменателя, например, R=(O+1)/(E+1). Взятие логарифма Log(R) проводили так, чтобы он отражал относительное увеличение представленности (Log(R)>0) или снижения представленности (Log(R)<0) каждого гена, и оказалось, что было недостаточно информации для количественного определения относительного увеличения/снижения представленности для этих генов. Среди генов с валидным вычислением Log(R), распределения Log(R) наносили на график для генов в наборе данных IP-MS, генов в наборе данных SILAC и генов ни из одного из наборов данных (фиг. 18C).
Анализ локализации исходного гена, связанный с фиг. 18D
[0702] С использованием того же показателя log(R), как описано выше, строили график распределения в соответствии с локализаций каждого гена-источника (фиг. 18D). Локализацию гена-источника определяли с использованием Uniprot (uniprot_sprot.dat).
Анализ данных экспрессии для класса II в данных RNA-Seq для единичных клеток, связанных с фиг. 19A
[0703] Данные RNA-Seq для единичных клеток получали из трех ранее опубликованных наборов данных, которые определяли профиль в образцах опухолей человека.
[0704] Первое исследование включало данные для кожной меланомы. Файл "GSE72056_melanoma_single_cell_revised_v2.txt" загружали из Gene Expression Omnibus (ncbi.nlm.nih.gov/geo/; номер доступа: GSE72056). Клетки с обозначением статуса опухоли "2" обрабатывались как опухолевые клетки, и клетки, обозначенные посредством обозначения статуса опухоли "1" и обозначением типа иммунных клеток, равным "1"-"6", обрабатывались как T-клетки, B-клетки, макрофаги, клетки эндотелия, фибробласты и NK, соответственно. Все другие клетки исключали. Данные естественным образом предоставляли в единицах log2(TPM/10+1) и, таким образом, их математически конвертировали до масштаба TPM. После достижения шкалы TPM данные для каждой клетки подвергали перенормализации до суммы 1000000 на протяжении набора кодирующих белок символов генов UCSC (кодирующие белок гены, которые не появлялись в матрице экспрессии, безусловно обрабатывались как имеющие нулевую экспрессию). Наконец, наблюдения для единичных клеток, соответствующие тому же типу клеток и тому же биоптату-источнику, усредняли для достижения оценок экспрессии на уровне типов клеток пациента.
[0705] Второе исследование включало данные для опухолей головы и шеи. Файл "GSE103322_HNSCC_all_data.txt" загружали из Gene Expression Omnibus (ncbi.nlm.nih.gov/geo/; номер доступа: GSE103322). Данные в этой таблице приведены в единицах log2(TPM/10+1); таким образом, величины математически конвертировали в единицы TPM. Как и в случае исследования меланомы, данные для каждой клетки подвергали перенормализации до суммы 1000000 на протяжении набора кодирующих белок символов генов UCSC, и наблюдения для единичных клеток, соответствующие одному и тому же типу клеток и одному и тому же исходному биоптату, усредняли. Данные, соответствующие биоптатам лимфатических узлов, исключали.
Третье исследование включало данные для немелкоклеточного рака легкого без лечения. Файлы "RawDataLung.таблица.rds" и "metadata.xlsx" загружали из ArrayExpress (ebi.ac.uk/arrayexpress/; номера доступа: E-MTAB-6149 и E-MTAB-6653). Данные (уже в единицах TPM), подвергали перенормализации до суммы 1000000 на протяжении набора кодирующих белок генов, как описано ранее. Наконец, наблюдения для единичных клеток, соответствующие одному и тому же типу клеток и одному и тому же биоптату-источнику, усредняли с получением оценки экспрессии на уровне типа клеток пациента. Проводили сходные исследования при раке ободочной и прямой кишки и раке яичника. Результаты указаны на фиг. 19A.
[0706] Для простоты типы клеток объединяли до более грубой степени детализации, чем первоначально описано в таблице 4.
Таблица 4
[0707] Уровни экспрессии HLA-DRB1 в пяти исследованиях нанесены на график на фиг. 19A.
Охарактеризация опухолевой против стромальной экспрессии класса II
[0708] Для определения относительного уровня экспрессии связывающего MHC класса II пептида, свойственного опухоли против стромы, были идентифицированные мутации в генах каскадов класса II у пациентов TCGA (называемые на основе ДНК), и для каждого пациента, имеющего мутацию класса II, проводили количественное определение относительной экспрессии мутантных и немутантных копий гена в соответствии с RNA-Seq. Кроме того, было предположено, что:
1. Мутантные чтения происходят из опухоли
2. Немутантные чтения происходят из стромы или аллелей дикого типа в опухоли
3. Опухоль сохраняет копию дикого типа с экспрессией, приблизительно равной экспрессии мутантной копии
[0709] Исходя из этого, бело определено, что для наблюдаемой доли мутантного аллеля f, доля экспрессии класса II, приписываемой опухоли, составляла приблизительно 2f и не более 100%. Три гена - CIITA, CD74 и CTSS - были отобраны в качестве центральных генов каскада класса II и оценены в отношении мутаций (не исключая синонимические мутации и мутации UTR) в TCGA (данные, загруженные с TumorPortal (tumorportal.org/): BRCA, CRC, HNSC, DLBCL, MM, LUAD; загруженные посредством пакетной загрузки TCGA (tcga-data.nci.nih.gov): CESC, LIHC, PAAD, PRAD, KIRP, TGCT, UCS; Synapse (synapse.org/#!Synapse:syn1729383): GBM, KIRC, LAML, UCEC, LUSC, OV, SKCM; или оригинальной публикации TCGA (cancergenome.nih.gov/publications): BLCA, KICH, STAD и THCA). Эти гены были отобраны на основе их известных ролей в экспрессии класса II и их высокой корреляции с HLA-DRB1 среди группы образцов 8500 GTEx. Другие гены с эквивалентной корреляцией с HLA-DRB1 (HLA-DRA1, HLA-DPA1, HLA-DQA1, HLA-DQB1 и HLA-DPB1) были исключены, поскольку их полиморфная природа делает их склонными к ложноположительным сигналам мутаций. Естественно, только небольшая доля пациентов имела мутацию в CIITA, CD74 или CTSS, и для некоторых типов опухолей отсутствовали доступные для анализа пациенты.
[0710] Последовательности из оригинального полноэкзомного секвенирования (WES) в формате Binary Sequence Alignment/Map (BAM) подвергали визуальной оценке (инструмент IGV) для подтверждения того, что мутация присутствовала в образце опухоли и не присутствовала в нормальном образце. Количества чтений мутантов против дикого типа получали из соответствующих данных RNA-Seq с использованием pysam. В целом, экспрессия HLA-DRB1 была определена на основе данных об экспрессии, загруженных с Genomic Data Commons (gdc.cancer.gov), которые были перенормализованы до суммы до 1000000 на протяжении набора кодирующих белок генов. Долю экспрессии HLA-DRB1, приписываемой опухоли (фиг. 19B), оценивали в качестве min(1,2f), где f представляет собой долю чтений RNA-Seq d CIITA, CD74 или CTSS, имеющих мутацию.
Оценка общей эффективности прогнозирования на натуральных донорских тканях
[0711] Периферическую кровь от семи здоровых доноров подвергали определению профиля с использованием DR-специфического антитела, как описано в разделе "выделение комплексов HLA-пептид на основе антител", выше. На основе этих результатов было определено два набора данных: один для аппроксимации многофакторной логистической регрессии, а другой для оценки эффективности прогнозирования для регрессий.
[0712] Первый набор данных конструировали с использованием алгоритма отбора наиболее подходящих последовательностей и ловушек, ранее описанного в отношении фиг. 4. В кратком изложении, это означает репрезентацию каждого вложенного множества посредством одного наиболее подходящего пептида (наиболее короткий пептид во вложенном множестве) и наложения совпадающих по длине ловушек на гены, так чтобы они минимально перекрывались с наиболее подходящими последовательностями и минимально друг с другом. Однако две важных детали отличаются от алгоритма, приведенного на фиг. 4. Во-первых, наиболее подходящие последовательности и ловушки выбирают из генов в части "настройки" (а не в "тестовой" части), и, во-вторых, ловушкам позволяли картироваться на генах, которые продемонстрировали ноль наиболее подходящих последовательностей. Модели логистической регрессии с показателями связывания MHC (из NetMHCIIpan или neonmhc2), а также другими вводными признаками (экспрессия, генное смещение и т.д.) обучали на этом наборе данных.
[0713] Второй набор данных (использованный для оценки), конструировали идентичным образом, за исключением того, что в нем использовались наиболее подходящие последовательности и ловушки, установленные из "тестовой" части. В дополнение к показателям связывания, следующие переменные использовались в подгруппе регрессий, как показано в таблице 5.
Таблица 5
[0714] Для оценки эффективности все n наиболее подходящих пептидов оценивали посредством данной логистической регрессии и оценивали вместе с набором из 499n ловушек (случайным образом отобранных без замены из полного набора ловушек). Наивысшие 0,2% пептидов в объединенной выборке обозначали как положительные результаты, и положительную прогностическую величину (PPV) вычисляли в качестве доли положительных результатов, которые представляли собой наиболее подходящие последовательности. Следует отметить, что, поскольку количество положительных результатов ограничивается количеством, равным наиболее подходящим последовательностям, отзыв в точности равен PPV в этом оценочном сценарии. Использование неизменного соотношения 1:499 среди аллелей помогает стабилизировать уровни эффективности, на которые в ином случае в высокой степени влияет количество наиболее подходящих последовательностей, наблюдаемых для каждого донора. Это считалось приемлемым, поскольку было предположено, что количество наиболее подходящих последовательностей в большей степени связано с экспериментальными условиями, чем со свойствами, присущим донорским клеткам. Соотношение 1:499 не отклоняется значительно от того, что использовалось бы, если бы понижение выборки не применялось.
[0715] В то время как предпочтительные варианты осуществления настоящего изобретения показаны и описаны в настоящем описании, специалистам в данной области будет очевидно, что такие варианты осуществления предоставлены только в качестве примера. Подразумевается, что изобретение не ограничивается конкретными примерами, приведенными в описании. В то время как изобретение описано применительно к вышеупомянутому описанию, подразумевается, что описание и иллюстрации вариантов осуществления в настоящем описании не следует истолковывать как ограничивающие. Многочисленные отклонения, изменения и замены без отклонения от изобретения станут понятными специалистам в данной области. Более того, должно быть понятно, что все аспекты изобретения не ограничиваются конкретными изображениями, конфигурациями или относительными соотношениями, указанными в настоящем описании, которые зависят от множества условий и переменных. Следует понимать, что при применении настоящего изобретения на практике можно использовать различные альтернативы вариантам осуществления изобретения, описанным в настоящем описании. Таким образом, предусматривается, что изобретение также охватывает любые такие альтернативы, модификации, отклонения или эквиваленты. Подразумевается, что приведенная формула определения определяет объем изобретения и что способы и структуры, входящие в объем этих пунктов формулы изобретения и их эквивалентов, охватываются ими.
ПРИМЕР 11: Высокопроизводительная идентификация и валидация эпитопов, связывающих аллель HLA класса II
[0716] В этом примере описан репрезентативный надежный высокопроизводительный способ с использованием времяразрешенного переноса энергии флуоресценции (TR-FRET) для идентификации и валидации новых связывающих аллель MHC-II пептидов. Анализ имеет несколько частей (1) трансфекцию клеток векторной конструкцией, подходящей для экспрессии и секреции цепей α и β MHC-II, имеющих флуоресцентную метку для анализа FRET, (2) очистка секретируемых белковых продуктов конструкции MHC-II, (3) проведение анализа пептидного обмена (фиг. 22A). На фиг. 22B и фиг. 23 далее проиллюстрирована конструкция и методика. Анализ, как описано в настоящем описании, обеспечивает быстрый и эффективной протокол детекции и валидации, поскольку он может не требовать стабильных клеточных линий и охватывает простые стратегии выделения. Кроме того, тетрамер или мультимер может использоваться для детекции антигенспецифических CD4 клеток, например, после введения спрогнозированных посредством neonmhc2 эпитопов in vivo, и индуцированный после этого иммунный ответ используют для подтверждения ответа CD4+ T-клеток.
Анализ CLIP-TR-FRET для идентификации высокоаффинных связывающих MHC класса II пептидов
[0717] В настоящем описании представлены иллюстративные векторы для экспрессии α- и β-цепей HLA класса II, контролируемой промотором CMV, в одной векторной конструкции, белковый продукт которой обеспечивает надлежащим образом свернутые пары цепей α и β. В надлежащем образом свернутой форме α- и β-цепей субъединица α1 и субъдиница β1 имеют форму димера, субъединица α1 и субъединица β1 образуют открытый акцепторный конец, способный акцептировать пептид, напоминающий физиологические конфигурации. Для цели данного анализа эти экспрессируемые с вектора белковые продукты HLA с надлежащим образом свернутой формой α- и β-цепей называются мономерами HLA. Экспрессирующая конструкция содержит линкер, один или более участков расщепления пептидов, секреторный сигнал, факторы димеризации, например c-Fos и Jun, связанные с мотивом биотинилирования (BAP) и 10X-His-метку (SEQ ID NO: 20). Для стабилизации мономеров и способствования секреции используют пептид-местозаполнитель. Пептид-местозаполнитель может представлять собой пептид CMV. Пептид-местозаполнитель может представлять собой пептид CLIP. Пептид-местозаполнитель может представлять собой пептид, идентифицированный посредством лигандома на основе MS для аллелей. Пептид-местозаполнитель может ковалентно связываться с пептидами HLA на открытом акцепторном конце пептида α1-β1.
[0718] Иллюстративная конструкция, используемая в рамках настоящего изобретения, кодирует пептид-местозаполнитель CLIP с частью расщепления тромбином между CLIP и β-цепью, как показано на фиг. 23 (верхняя панель). При трансфекции и культивировании трансфицированных клеток, так чтобы они достигали оптимального роста, супернатант (среду) клеточной культуры, содержавший секретируемые белки (мономеры), собирали и пропускали через никелевые (Ni2+) колонки для очистки (фиг. 24). Уровни экспрессии и очистку изучали посредством окрашивания кумасси. 28-мерный пептид CLIP остается связанным с β-цепью, которая расщепляется посредством обработки тромбином (фиг. 24) и после этого может вытесняться посредством конкуренции с тестируемым связывающим HLA класса II пептидом (например, кандидат). Тестируемый пептид, который успешно вытесняет пептид CLIP, объяснимо представляет собой когнатный пептид для связывания с гетеродимером MHC-II конструкции, на основе способности вытеснять пептид CLIP при определении по его IC50. Для конкурентной реакции вытеснения, как описано выше, может использоваться тестируемый пептид de novo, который затем может быть идентифицирован посредством масс-спектрометрии (MS).
[0719] Большую коллекцию гетеродимерных конструкций HLA-DR получали с использованием пептидов-местозаполнителей CLIP, которые успешно секретировались, и проводили анализ пептидного обмена.
[0720] Было выявлено, что пептид-местозаполнитель CLIP, происходящий из CD74, имеет значительный эффект на секрецию мономеров HLA класса II. Как правило, в качестве последовательности местозаполнителя использовали отредактированный канонический пептид CLIP, имеющий последовательность CD74 PVSKMRMATPLLMQA (SEQ ID NO: 1) (обозначаемый как CLIP0 на фиг. 25A-25C). Однако наблюдалось, что некоторые пептиды HLA-DR, например, DRB1*12:01 и DRB1*13:02 имели низкий выход в случае канонического пептида (таблица 6). Было обнаружено, что определенные димеры аллелей HLA DRB имеют более длинную связывающую последовательность, охватывающую все или части аминокислот в последовательности: LPKPPKPVSKMRMATPLLMQALPM (SEQ ID NO: 21) (CLIP1) (фиг. 25A). Действительно, использование последовательности CLIP1 вместо последовательности CLIP0 в случае DRB1*12:01 и DRB1*13:02 повышало выход секреции димеров HLA (фиг. 25B-25C).
Таблица 6
Скрининг пептидов de novo посредством последовательного анализа пептидного обмена с использованием STII-TR-FRET
[0721] Пептиды можно подвергать скринингу de novo с использованием анализа, вовлекающего экспрессию мономерных белков HLA, описанных выше, в клеточных линиях, таких как клетки Expi293, собирать и очищать из супернатанта, и подвергать анализу пептидного обмена. Связывающие HLA класса II пептиды, спрогнозированные посредством алгоритмов прогнозирования, тестировали с использованием анализа пептидного обмена. Анализ пептидного обмена проводили с использованием способа, вовлекающего флуоресцентную поляризацию. Например, любой флуорофор может использоваться либо для мечения пептида-местозаполнителя, либо для мечения тестового пептида, либо для мечения обоих из них с использованием двух различных флуорофоров. Для количественной оценки реакции вытеснения может регистрироваться изменение флуоресценции либо в результате утраты связанного пептида-местозаполнителя, который предварительно мечен флуорофором, либо в результате испускания флуоресценции высвободившегося флуорофора, который ранее гасился биохимическими реакциями в его связанной с HLA форме. Альтернативно, может быть зарегистрировано вытеснение нефлуоресцентного пептида-местозаполнителя меченым флуоресцентным пептидом для количественного определения реакции вытеснения. В иллюстративном анализе использовали FITC-меченный пептид-местозаполнитель CLIP для вытеснения существующего ковалентно связанного пептида, такого как пептид CMV. FITC-меченный пептид, когда он связан с HLA, индуцирует высокую поляризацию. Когда FITC-пептид-местозаполнитель титруют с тестовым пептидом, тестовый пептид вытесняет FITC-CLIP, что приводит к снижению флуоресценции.
[0722] Анализ пептидного обмена также можно проводить с использованием технологии времяразрешенного FRET (TR-FRET) вместо флуоресцентной поляризации, как описано в настоящем описании. В иллюстративном анализе TR-FRET, описанном в настоящем описании, клетки трансфицировали мономерной конструкцией HLA, имеющей пептид-местозаполнитель, который содержал часть Streptag II (STII). Детекцию STII проводили с использованием меченного посредством Alexa-647 антитела к STII. В то же время детекцию His-метки, связанной с Jun-концом мономерной конструкции, описанной в этом примере ранее, которая находится вблизи конца α2-β2 пептидов HLA, проводили посредством соединения европия III (Eu), связанного с антителом против His (фиг. 26A). Комплекс Eu выступает в качестве донора энергии, в то время как Alexa647 выступает в качестве акцептора в реакции FRET, когда пептид-местозаполнитель остается связанным с мономером HLA. Когда тестируемый пептид вытеснял местозаполнитель STII-CLIP, пептид Alexa-647-αSTII высвобождался, но более не мог быть обнаружено по флуоресценции. Было обнаружено, что TR-FRET является более надежным, чем флуоресцентная поляризация. Кроме того, этот анализ имел значительно сниженный фоновый сигнал (флуоресцентные данные представлены на фиг. 26B-26E). Анализ обеспечивает высокопроизводительную платформу идентификации для пар HLA-пептид. Как показано в таблице 7 ниже, тестируемые пептиды (или пептиды-кандидаты) с P-156 по P-191, демонстрировали широкий диапазон способности к вытеснению и аффинности связывания при определении по вычисленной IC50 при каждом прогоне. Более низкая IC50 демонстрирует более высокую способность к вытеснению и более высокую связывающую способность.
Таблица 7
Прогон 1
Прогон 2
(нМ)
Валидация пептидного обмена с использованием дифференциальной сканирующей флуориметрии (DSF)
[0723] В этом способе проводится высокопроизводительный анализ для скрининга пептидов, которые могут связываться с конкретным аллелем HLA, а также определяют интенсивность связывания пептида с димером HLA (фиг. 26F). В этом анализе используется флуоресцентный зонд, который связывается с гидрофобными остатками белка и, таким образом может связываться с аллелями MHC только когда аллели диссоциируют друг от друга с использованием нагревания. Когда димер MHC класса II связывает распознаваемый им пептид, димеры удерживаются вместе в его димерной форме. Когда нагревание применяют к димеру MHC-пептиду в связанной форме, пептиды со слабым связыванием диссоциируют быстрее из белкового димера MHC класса II, позволяя флуорофору связываться с диссоциированными альфа- и бета-цепями MHC и вызывая высокую флуоресценцию. Флуоресценцию регистрируют в качестве функции температуры. Репрезентативные кривые плавления представлены на фиг. 26F. Кривые плавления можно сравнивать для определения сильно связывающихся структур (детекция флуоресценции при более высокой температуре) и слабо связывающихся структур (детекция флуоресценции при более низкой температуре).
[0724] Использование растворимого HLA-DM (HLA-sDM) в качестве катализатора для пептидного обмена с MHC класса II: HLA-DM является природным шапероном и катализатором пептидного обмена для молекул HLA-DR, -DP и -DQ. Он является интегральным мембраным белком и встречается в качестве гетеродимера полипептидных цепей альфа и бета (DMA и DMB). Пептидный обмен, как описано в этом разделе, проводят с использованием растворимой формы HLA-DM (например, белок HLA-sDM) в качестве шаперона для обмена HLA-DR, -DP и -DQ. Белок HLA-sDM продуцируют посредством временной трансфекции в клетках Expi-CHO, как показано на фиг. 26G. В кратком изложении, конструируют рекомбинантную конструкцию HLA-sDM, как графически представлено на верхней половине фиг. 26G. Рекомбинантная конструкция HLA-sDM содержит конститутивный промотор CMV выше лидерной последовательности и функционально связанный с промотором. Лидерная последовательность способствует секреции продукта (секреторный сигнал). На 3’-конце лидерной последовательности внесена кодирующая последовательность для эктодомена бета-цепи HLA-DM (и она лишена трансмембранного домена). Последовательность, кодирующая мотив биотинилирования (BAP), лигирована с 3’-стороны от кодирующей бета-цепь последовательности. Последовательность, кодирующую экодомен альфа-цепи HLA-DM (и лишенную трансмембранного домена) помещают с секреторной последовательностью (лидерной последовательностью) на 5’-конце, и она отделена от последовательности BAP встроенной последовательностью рибосомального пропускания. Последовательность альфа-цепи HLA-DM лигирована на 3’-конце с 10X HIS-меткой (SEQ ID NO: 20). После образования гетеродимерный HLA-sDM секретируется наружу клетки. Когда эту конструкцию экспрессируют в клетках Expi-CHO, белок HLA-sDM секретируется в культуральную среду.
[0725] Клетки Expi-CHO трансфицировали плазмидным вектором, экспрессирующим конструкцию HLA-sDM, и культивировали в течение приблизительно 14 суток. Белок секретировался в культуральную среду в ходе культивирования. Белок HLA-sDM очищали из культуры в процессе, очень сходном с очисткой белков MHC-II. Пептидный обмен MHC-II можно проводить эффективно с кислотой и HLA-sDM или без кислоты, и с октилглюкозидом. Для оценки пептидного обмена проводили эксклюзионную хроматографию, результаты были такими, как показано на фиг. 26H. Все анализы пептидного обмена проводили с использованием HLA-sDM или октилглюкозида в качестве катализатора.
Репертуар тетрамеров (или мультимеров) HLA-класса II
[0726] Большой репертуар тетрамеров HLA класса II получали для тестирования связывания эпитоп:HLA и кинетики диссоциации в биохимическом анализе. Эти тетрамеры класса II, полученные таким образом, используются для анализа связывания и презентации пептидов. Например, тетрамеры использовали в анализе пептидного обмена. Как показано на фиг. 27A было получено 12 тетрамеров, и их хранили в концентрации более 15 мг/мл; шесть тетрамеров и четыре в концентрации <5 мг/мл. Тетрамеры HLA использовали в проточной цитометрии для идентификации реактивных к неоантигенам CD4+ T-клеток. Эпитоп вируса гриппа (HA) и эпитопы ВИЧ тестировали в отношении распознавания T-клетками, когда они презентировались тетрамерами HLA (фиг. 27E).
[0727] На фиг. 27B-27D представлены различные подгруппы тетрамеров HLA класса II, которые были получены и очищены. Как показано на фиг. 27B, большой репертуар тетрамеров гетеродимеров DRB1 был сконструирован и очищен в концентрации более 15 мг/л. На фиг. 27C и 27D обобщенно представлен охват конструкций для аллелей MHC класса II человека, которые были получены и валидированы для анализов связывания пептидов на флуоресцентной основе. В таблице 8A, таблице 8B и таблице 8C приведены перечни полученных аллельных тетрамеров с соответствующими концентрациями выхода секреции очищенного продукта.
Таблица 8A
Таблица 8B
Таблица 8C
[0728] Процесс получения тетрамеров MHC-II, кроме того, включает аллели DRB3, 4 и 5, и аллели DP и DQ.
Валидация пептидного обмена с использованием флуоресцентной поляризации (FP)
[0729] Флуоресцентную поляризационную микроскопию использовали в анализе для отличения пептида, связанного с белками MHC класса II, от свободных пептидов. Флуорецентно меченный пептид-местозаполнитель, когда он связан с димером MHC класса II, обеспечивает высокополяризованный свет посредством флуоресцентной поляризационной (FP) микроскопии, по сравнению с его высвободившейся формой, когда не меченный флуорофором конкурирующий эпитоп остается связанным с MHC класса II посредством вытеснения пептида-местозаполнителя. На фиг. 28A представлен принцип посредством графического представления. В кратком изложении, анализ проводят посредством следующего обобщенного способа, и его варианты либо указаны в соответствующем описании, либо хорошо понятны специалисту в данной области.
[0730] Реагенты, как описано в таблице 9 (ниже) собирают в реакционной пробирке (например, 1,5-мл пробирке Eppendorf), хорошо перемешивают и инкубируют при 37°C в течение 2 часов. К смеси добавляют 25 мл 10X PBS в конце времени инкубации для нейтрализации реакции пептидного обмена.
Таблица 9
[0731] Детекцию осуществившего обмен пептида проводят, например, путем окрашивания; или его хранят при -80°C посредством быстрого замораживания в жидком азоте для дальнейшей оценки.
[0732] На фиг. 28B и 28C представлен обзор разработки анализа для HLA DRB1*01:01 с использованием FP и различных использованных условий. В некоторых примерах определяли эффект pH на анализ. В кратком изложении, в клетках экспрессируют аллели как полноразмерных, так и растворимых белков. Мембраносвязанную полноразмерную форму белка аллеля собирают посредством пермеабилизации мембраны, в то время как секретируемую форму собирают из клеточного супернатанта. Собранные белки аллелей HLA класса II очищают, пропуская их через никелевые колонки (Ni2+). В некоторых примерах оценивали эффект детергента (1% октилглюкозид против 1,6% NP40) в отношении пермеабилизации мембраны для сбора полноразмерных белков аллелей MHCII. В некоторых примерах оценивали индивидуально эффект температуры или использованного зонда, или способов очистки, или формата мишени (фиг. 28B).
[0733] Оценивали эффект либо способа очистки с использованием конформационно-специфического антитела L243, либо способа очистки с His-меткой. Результаты представлены на фиг. 28D. Каждая точка данных представлена в таблице слева, и она представлена в качестве единичной точки на графике справа. Точки на графике находятся приблизительно под углом 45 градусов к любой из оси и имеет величину r 0,9621, которая указывает на то, что величины IC50 из обоих способов очистки согласуются друг с другом. Также показано, что ранговый порядок эффективности пептидов не изменяется между способами очистки.
[0734] Проводили оценку эффекта выбора белков HLA класса II в растворимой форме (sDR1) против полноразмерной формы (fDR1), и на фиг. 28E показано, что выбор формата мишени не влияет на эффективность пептида. Слева представлены средние величины IC50 из экспериментов с использованием формы sDR1 или fDR1. Эти данные нанесены на график, представленный справа. Точки данных, в каждом случае отображенные единичной точкой, приблизительно находятся под углом 45 градусов к любой из осей и имеют величину r 0,9365, что указывает на то, что величины IC50 для обеих использованных форм хорошо коррелируют друг с другом. Также на нем показано, что ранговый порядок эффективности пептидов не изменяется между способами очистки.
[0735] На фиг. 28F представлено графическое изображение иллюстративного способа оценки спрогнозированных пептидов посредством neonmhc2 и NetMHCIIpan в анализе связывания реальных пептидов. Для оценки спрогнозированных посредством Neonmhc2 и NetMHCIIpan пептидов в анализе связывания реальных пептидов использовали анализ флуоресцентной поляризации. Для анализа 60 нМ расщепленный тромбином растворимый HLA-DRB1*15:01 инкубировали с FITC-меченным суперсвязывающим пептидом-зондом (PVVHFFK(FITC)NIVTPRTPPY (SEQ ID NO: 24)) (10 нМ на анализ) и анализируемым пептидом в течение 5 часов при 37°C в буфере для анализа (pH 5,2). Исследовали флуоресцентную поляризацию, из которой вычисляли % вытеснение зонда. Как показано на фиг. 28G, ингибирование суперсвязывающего флуоресцентного пептида было пропорционально концентрации спрогнозированного пептида, что указывает на высокую специфичность анализа. Между эффективностью пептидов, спрогнозированных посредством Neonmhc2 и NetMHCIIpan, наблюдались поразительные различия. В случае спрогнозированных посредством Neonmhc2 пептидов большее количество пептидов связывались на положительном уровне и с более высокой степенью ингибирования; в то время как спрогнозированные посредством NetMHCIIpan пептиды в целом имели низкую эффективность по сравнению с пептидами Neonmhc2. На фиг. 28H представлено обобщение оценки спрогнозированных посредством neonmhc2 пептидов в анализе связывания. Как показано посредством круговых диаграмм, среди дважды негативных пептидов (пептиды, которые не были спрогнозированы ни одним из NetMHCIIpan или Neonmhc2) только 5% оказались связывающимися и 95% оказались не связывающимися. Из спрогнозированных посредством NetMHCII пептидов 40% были связывающимися посредством детекции с использованием флуоресцентной поляризации в анализе вытеснения зонда, и было обнаружено, что из спрогнозированных посредством Neonmhc2 пептидов в качестве связывающихся пептидов 100% были реально связывающимися в анализе вытеснения зонда.
[0736] FITC-меченные зонды получали путем изучения ранее опубликованных пептидов, для которых было показано Sette et al., что они связывают определенные аллели. Затем эти пептидные последовательности анализировали с использованием спрогнозированного центра связывания класса II для идентификации минимального 9-мерного центра пептида и якорных остатков. Затем эту информацию рассматривали при выборе положения остатка для замены на лизин и мечения посредством FITC. Например, в таблице ниже (таблица 10) приведены последовательности, как описано Sette et al. (Sidney J, Southwood S, Moore C, et al. Measurement of MHC/peptide interactions by gel filtration or monoclonal antibody capture. Curr Protoc Immunol. 2013;Chapter 18:Unit-18.3. doi:10.1002/0471142735.im1803s100) (далее «последовательности Sette»). Спрогнозированный центр связывания каждого пептида для класса II подчеркнут в контексте конкретного аллеля. Полужирным шрифтом указаны якорные положения, которые были идентифицированы в результате усовершенствования эпитопов. В некоторых случаях, одна и та же пептидная последовательность может использоваться для различных аллелей.
Таблица 10
[0737] На основе положения остатков, как описано выше, был выбран внутренний лизин для конъюгации FITC посредством фокусирования на положениях в связывающем центре (подчеркнуты), которые не выделены синим цветом - эти положения выделены красным цветом в качестве положений, подходящих для конъюгации с FITC. Для последовательностей, которые не имели внутреннего лизина для конъюгации с FITC, использовали подход изучения вручную, где проводили сравнение связывающего мотива аллеля с пептидной последовательностью, и было выбрано положение для внутренней замены на лизин для пептидов DRB1*09:01 и DRB1*03:01 (см. таблицу выше). Более конкретно, остаток лейцина для DRB1*09:01 и остаток аргинина для DRB1*03:01 были замещены остатками лизина, чтобы обеспечить конъюгацию с FITC. Эта стратегия замены была основана на установленных посредством MAPTAC мотивах, где применялась идентификация вручную положений без высоких предпочтений аминокислот (также в середине спрогнозированной посредством neonmhc2 9-мерной центральной части), поскольку конъюгированный флуорофор может с большей вероятностью испускать поляризованный свет в связанном состоянии (т.е. более ограниченном движении флуорофора).
ПРИМЕР 12: Связывание HLA класса II и правила процессинга для идентификации антигенов злокачественной опухоли в качестве терапевтических мишеней
[0738] Накапливающиеся данные указывают на то, что CD4+ T-клетки могут распознавать специфические для злокачественной опухоли антигены и контролировать рост опухоли. Однако остается трудным предсказать антигены, которые будут презентироваться молекулами лейкоцитарного антигена класса II человека (HLA класса II) - препятствуя попыткам оптимального нацеливания на них терапевтически. Препятствия включают неточное прогнозирование связывания пептидов и неразгаданную сложность каскада HLA класса II. В этом примере описана усовершенствованная технология для обнаружения связывающих HLA класса II мотивов. Кроме того, в настоящем описании описан всесторонний анализ опухолевых лигандомов, проведенный для изучения правил процессинга в отношении микроокружении опухоли (TME).
[0739] Проводили определение профиля для 40 аллелей HLA класса II, и было показано, что связывающие мотивы являются в высокой степени чувствительными к HLA-DM, загружающему пептиды шаперону. Было обнаружено, что внутриопухолевая презентация HLA класса II определяется профессиональными антигенпредставляющими клетками (APC), а не злокачественными клетками. Объединяя эти наблюдения, были разработаны алгоритмы, как описано в настоящем описании, которые точно прогнозируют лигандомы APC, включая пептиды из фагоцитированных злокачественных клеток. Эти инструменты и биологические знания могут улучшить направленные на HLA класса II средства терапии злокачественной опухоли.
[0740] Перспективный новый класс терапевтических средств направлен на лечение злокачественной опухоли путем индукции T-клеточных ответов против антигенов злокачественной опухоли и подвергнутых соматической мутации последовательностей, называемых неоантигенами. В настоящее время эти усилия сфокусированы в основном на индукции ответов CD8+ T-клеток на лиганды, презентируемые HLA класса I (HLA класса I). Однако несколько последних исследований показали, что CD4+ T-клетки также могут распознавать презентируемые посредством HLA класса II лиганды и участвовать в контроле опухоли. В вакцинах против злокачественной опухоли и других иммунотерапевтических средствах в идеале используется направление ответов CD4+ T-клеток, однако текущие усилия полностью упускают прогнозирование антигена HLA класса II, поскольку точность современных инструментов прогнозирования является недостаточной.
[0741] Ключевым фактором, препятствующим точной идентификации антигенов злокачественной опухоли HLA класса II, является недоступность исчерпывающих высококачественных данных для изучения правил связывания пептидов. Необходимы данные для трех высокополиморфных канонических локусов HLA класса II: HLA-DR, -DP и -DQ, где каждый аллельный вариант демонстрирует характерное предпочтение связывания. Широко используемым способом определения связывающих пептид мотивов является биохимический анализ, который измеряет аффинность единичного пептида в отсутствии физиологических шаперонов, таких как HLA-DM. Охват измеренных данных аффинности ограничивается распространенными аллелями HLA-DR, и даже для этих аллелей точность прогнозирования значительно отстает от HLA класса I. В принципе, лигандомика на основе масс-спектрометрии (MS) должна обеспечить усовершенствованное прогнозирование путем обеспечения масштабируемости и эндогенных условий нагрузки пептидами. Тем не менее, натуральные лигандомы являются мультиаллельными, и информация о картировании пептидов на аллелях, требуемая для получения точных обучающих данных, скрыта. В решении этой проблемы для HLA класса I произошел прогресс с использованием как обратной свертки, так и моноаллельных по HLA класса II клеточных линий; наборы данных для лигандома моноаллельного HLA класса II были получены с использованием низкопроизводительных трансгенных моделей на мышах с дефицитными по HLA класса II клеточными линиями или клеточными линиями, которые имеют гомозиготный аллель HLA-DR.
[0742] Другой проблемой является неясность, какие опухолевые антигены наиболее вероятно подпадут в каскад презентации HLA класса II. В последних исследованиях на основе MS изучались лигандомы HLA класса II для образцов опухоли, но они не были направлены на то, презентируют ли профессиональные APC или злокачественные клетки терапевтически значимые антигены HLA класса II. Более того, в настоящее время неизвестно, зависит ли процессинг посредством HLA класса II опухолевых антигенов в основном от фагоцитоза или аутофагии. В зависимости от того, какой путь преобладает в соответствующем типе клеток, могут существовать значительные различия с точки зрения того, какие белки являются предпочтительными в качестве источников для пептидных лигандов HLA класса II. Проблему усложняет то, что не существует системного подхода для определения того, какие области в белках наиболее вероятно будут продуцировать лиганды HLA класса II, даже несмотря на то, что преобладающая теория состоит в том, что признаки белковой последовательности должны влиять на потенциал HLA класса II к процессингу.
[0743] Для исследования правил процессинга и презентации антигенов HLA клетки II для терапевтического нацеливания, следовали двухстороннему подходу, состоящему в i) усовершенствовании прогнозирования связывания пептидов и ii) определении процессинга и презентации лигандов HLA класса II в TME. Для изучения аллелеспецифического связывания пептидов был разработан масштабируемый моноаллельный процесс определения профиля лигандома HLA, называемый MAPTAC™ (моноаллельная очистка с мечеными аллельными конструкциями (Mono-Allelic Purification with Tagged Allele Constructs)), в котором используется MS для секвенирования эндогенно презентируемых лигандов HLA класса II. MAPTAC™ позволяет отчетливо разделить пептид-связывающие мотивы для 40 аллелей HLA класса II и обучить алгоритмы прогнозирования связывания, которые могут точно идентифицировать иммуногенные вирусные эпитопы и неоантигены. Для усовершенствования прогнозирования процессинга для HLA класса II анализировали образцы опухоли, установив профессиональные APC в качестве основного источника внутриопухолевой экспрессии HLA класса II и определив набор генов и генных областей, предпочтительно процессируемых этими клетками. Затем было продемонстрировано, что алгоритмы, которые интегрируют признаки связывания и процессинга, могут прогнозировать естественные лигандомы APC и, более важно, подгруппу лигандов HLA класса II, происходящих из подвергнутых эндоцитозу злокачественных клеток. Эти достижения в понимании правил процессинга и презентации терапевтически значимых антигенов HLA класса II обеспечат средства терапии, которые нацелены на привлечение ответов CD4+ T-клеток.
МЕТОДИКИ ЭКСПЕРИМЕНТА
Конструирование конструкции MAPTAC™ и культивирование клеток
[0744] Для HLA класса I α-цепь подвергали слиянию с C-концевым линкером GSG, за которым следовала последовательность биотин-акцепторного пептида (BAP), стоп-кодон и вариабельный ДНК-штрихкод, и клонировали в вектор pSF Lenti (Oxford Genetics). Аналогично конструкции HLA класса II клонировали в pSF Lenti, и они составляли из последовательности β-цепи с той же последовательностью линкер-BAP, слитой а C-конце, за которой следовал другой короткий линкер GSG, последовательность рибосомального пропускания F2A, последовательность α-цепи с C-концевой HA-меткой, стоп-кодон и вариабельный ДНК-штрихкод. Конструкции MAPTAC™ трансфицировали или трансдуцировали в клетки Expi293, HEK293T, A375, HeLa, KG-1, K562 и B721.221.
Протоколы выделения HLA-пептидов
[0745] Быстрозамороженные клеточные осадки, содержащие 50×106 клеток, экспрессирующих BAP-меченный HLA, размораживали на льду в течение 20 минут и осторожно лизировали посредством пипетирования вручную в 1,2 мл холодного лизирующего буфера. После очистки от ДНК, РНК и клеточного дебриса супернатанты переносили в новые 1,5-мл пробирки и BAP-меченный HLA биотинилировали посредством инкубации при комнатной температуре в течение 10 минут с 0,56 мкМ биотином, 1 мМ ATP, и 3 мкМ BirA. Биотинилированные лизаты инкубировали с 200 мкл смолы с нейтравидином при 4°C в течение 30 минут для аффинного обогащения биотинилированных комплексов HLA-пептид. После промывания связанную с HLA смолу осаждали центрифугированием при 1500×g при 4°C в течение одной минуты и хранили при -80°C или сразу подвергали элюированию HLA-пептида и обессоливанию с использованием твердофазной экстракции Sep-Pak. Для определения профиля эндогенных лигандомов HLA класса II материалов от здоровых доноров, комплексы HLA-пептид выделяли с использованием созданного в собственной лаборатории антитела против HLA-DR L243 или с использованием коммерчески доступного антитела TAL 1B5.
Секвенирование HLA-пептид посредством тандемной масс-спектрометрии
[0746] Во всех анализах nanoLC-ESI-MS/MS использовались одни и те же условиях разделения LC, параметры устройства и способы анализа данных. В кратком изложении, образцы подвергали хроматографическому разделению с использованием Proxeon Easy NanoLC 1200, оборудованного колонкой PicoFrit, которую самостоятельно набивали гранулами C18 Reprosil, и нагревали при 60°C. В ходе зависимого от данных получения результатов, элюированные пептиды подавали на масс-спектрометр Orbitrap Fusion Lumos, оборудованный источником ионов Nanospray Flex Ion source. Спектры масс интерпретировали с использованием пакета программ Spectrum Mill v6.0 pre-Release. Идентифицированные пептиды, которые удовлетворяли оценке PSM FDR <1%, фильтровали в отношении примесей посредством удаления всех пептидов, отнесенных к 264 распространенным белкам-примесям в эталонной базе данных и путем удаления пептидов, идентифицированных в аффинном анализе соосаждения MAPTAC™ для отрицательного контроля. Кроме того, все пептиды, которые картировались на трипсинизированном продукте расщепления in silico эталонной базы данных, удаляли для учета переноса из трипсинизированного образца. Исходные наборы данных масс-спектрометрии депонированы в MassIVE после получения (massive.ucsd.edu).
Машинно-обучаемые подходы для связывающих мотивов и прогнозирования связывания
[0747] Для каждого аллеля систему сверточных нейронных сетей обучали для отличения пептидов MAPTAC™ от перемешанных ловушек. Каждая сеть включала два ReLU-активированных сверточных слоя, каждый с 50 фильтрами шириной 6. Максимальная и средняя активация на фильтр на слой направлялись в конечный плотный слой с сигмоидальной активацией. Регуляризация достигалась посредством L2-нормализации с 20% пространственным исключением после каждого сверточного слоя, и ранней остановки, и настраивалась по аллелям в соответствии с удержанной частью неизбыточных пептидов (~12,5%). При сопоставлении эффективности прогнозы NetMHCIIpan-v3.1 вычислялись в качестве 15-мера с максимальной оценки в каждом пептиде запроса, и этот подход имел единообразно лучшую эффективность, чем нативное прогнозирование посредством NetMHCIIpan-v3.1.
Анализ индукции CD4+ T-клеток
[0748] PBMC сокультивировали с обработанными пептидами mDC в соотношении 1:10 с всего 3 стимуляциями. Затем индуцированные T-клетки метили уникальным двухцветным штрихкодом, как описано ранее, и культивировали в течение ночи в соотношении 1:10 с обработанными пептидам и созревшими аутологичными mDC. Затем клетки оценивали в отношении продуцирования IFN-γ в ответ на пептид посредством проточной цитометрии. Индуцированные образцы, которые положительно отвечали на пептид, представляли собой образцы, в которых индуцировалось продуцирование IFN-γ на 3% выше, чем в контроле без пептида.
Эндоцитоз посредством APC меченных SILAC опухолевых клеток
[0749] Клетки K562 (ATCC, Manassas, VA) выращивали на протяжении 5 удвоений в среде RPMI для SILAC (ThermoFisher), содержащей меченные тяжелыми изотопами аминокислоты, L-лизин 2HCl 13C6 15N2 (Life Technologies) и L-лейцин 13C6 (Life Technologies). Происходящие из моноцитов дендритные клетки (mDC) сокультивировали в соотношении 1:3 либо в течение ночи с УФ-обработанными клетками K562, либо в течение 5 ч с лизатом, полученным после обработки HOCl. Клетки собирали, осаждали и быстро замораживали в жидком азоте для протеомного анализа.
РЕЗУЛЬТАТЫ
MAPTAC™: Масштабируемая платформа для моноаллельного определения профиля лигандов HLA класса II
[0750] Современное знание связывающих HLA класса II мотивов основано в основном на данных, полученных с использованием двух биохимических анализов связывания. В одном таком подходе анализируемый пептид и меченный радиоактивной меткой конкурирующий пептид совместно инкубируют с экстрактами HLA клеточного происхождения для определения IC50. В другом подходе специфическое для конформации антитело определяет долю HLA, связанного с анализируемым пептидом, для определения EC50. Данные этих анализов объединены в базе данных Immune Epitope Database (IEDB) и используются для обучения алгоритмов для прогнозирования HLA класса II, таких как NetMHCIIpan. Пять наиболее распространенных аллелей HLA-DRB1 у европиоидов хорошо представлены в IEDB (3326-8967 пептидов для каждого), хотя только приблизительно 29% из них представляют собой структуры с сильным связыванием (аффинность <100 нМ), и 85% пептидов IEDB в целом являются точными 15-мерами (фиг. 12B, фиг. 12E). Аллели HLA-DP и HLA-DQ и аллели HLA-DR не европиоидов (например, HLA-DRB1*15:02) представлены значительно меньшим количеством данных.
[0751] Для получения высококачественного набора данных с аллельной широтой, представляющей многообразную популяцию пациентов, была разработана MAPTAC™, технология, которая позволяет эффективное выделение пептидов HLA класса II, связывающихся с единичным аллелем, для идентификации на основе MS (фиг. 11A). Альфа- и бета-цепи выбранного гетеродимера HLA класса II кодируются генетической конструкцией с последовательностью биотин-акцепторного пептида (BAP), находящейся на C-конце бета-цепи. Поскольку HLA-DRA функционально инвариантен, MAPTAC™ обеспечивает моноаллельные результаты HLA-DR независимо от потенциального образования пар между экзогенной бета-цепью и эндогенной альфа-цепью. Для HLA-DP и HLA-DQ, которые демонстрируют ограниченный набор функциональных вариантов альфа-цепи, выбирают клеточную линию, имеющую совпадающие или не экспрессируемые аллели альфа-цепи. Важно, что этот подход также работает для HLA класса I с BAP-меткой, присоединенной к тяжелой цепи HLA класса I.
[0752] 48-часовая трансфекция достигала устойчивой экспрессии конструкции MAPTAC™ (фиг. 12C) с нормальными уровнями презентации на клеточной поверхности (фиг. 34A и 34B). Это было продемонстрировано в 7 различных клеточных линиях (expi293, A375, KG-1, K562, HeLa, HEK293 и B72.221) и для 40 аллелей HLA класса II, обеспечивая данные для всех трех канонических локусов HLA класса II: HLA-DR, -DP и -DQ. Среднее количество идентификаций уникальный пептидов на реплику (~50 миллионов клеток), которые проходили фильтры контроля качества, находились в диапазоне от 236 до 2580 среди аллелей (фиг. 29), со срединным количеством 1319 пептидов. Для увеличения глубины данных использовали несколько модификаций процесса, включая сверхэкспрессию HLA-DM, и восстановление и алкилирование пептидов. Только небольшой процент наиболее подходящих последовательностей MS соответствовал известным примесям, трипсинизированным пептидам и имитирующим трансфекциям (пустая плазмида) (фиг. 11B и фиг. 29). Распределение длин для пептидов HLA класса I и HLA класса II MAPTAC™ соответствуют тем, которые наблюдались в предшествующих исследованиях MS с использованием соосаждения на основе антител (фиг. 11C).
[0753] Среди пептидов HLA класса II MAPTAC™ большинство аминокислот были представлены на уровнях, согласующихся с исходными частотами в протеоме (фиг. 12D и фиг. 12F). Исключения включали C, M и W, которые имели сниженную представленность на 85%, 34% и 42%, соответственно, что согласуется с предшествующими исследованиями на основе MS для пептидов HLA класса II. Восстановление и алкилирование пептидов HLA класса II практически утраивало частоту C, хотя она была все еще представлена на сниженном уровне относительно протеома (фиг. 12F). Снижение представленности C, M и W не наблюдали в совпадающих по аллелю высокоаффинных пептидах (<100 нМ) из IEDB. Напротив, связывающие IEDB структуры демонстрировали уменьшение представленности D (-39%) и E (-37%), а также увеличение представленности M (+65%) по сравнению с не связывающими структурами согласно IEDB (>5000 нМ). Таким образом, MAPTAC™ демонстрирует определенное смещение, которое согласуется с со смещением, наблюдаемым для других технологий.
MAPTAC™ определяет мотивы, связывающие пептиды HLA класса II
[0754] MAPTAC™ использовали для определения аллелеспецифических связывающих HLA класса II мотивов. Проводили определение профиля 40 аллелей HLA класса II, 15 из которых ранее не были охарактеризованы (<30 пептиды c аффинностью <100 нМ в IEDB), включая аллели, общие в популяциях не европиоидов (DRB1*12:02, DRB1*15:03 и DRB1*04:07). Поскольку пептиды HLA класса II могут быть более длинными, чем количество остатков в связывающей бороздке, не является абсолютно очевидным, какая часть каждого пептида взаимодействует с HLA ("центральная часть"), а какая является выступающей; однако, определение связывающего центра является ключевым для охарактеризации связывающих мотивов. Для идентификации связывающего центра проводили выравнивание пептидов с консенсусным связывающим центром с использованием инструмента GibbsCluster-2.0, который использует алгоритм ожидания-максимизации для итеративного определения уровня связывания для каждого пептида и повторного изучения мотива связывания среди пептидов. За небольшими исключениями, мотивы связывающего центра для распространенных аллелей HLA-DR продемонстрировали высокую согласованность с мотивами на основе IEDB (фиг. 35). Выявленные посредством MAPTAC™ пептиды не всегда демонстрировали высокие показатели NetMHCIIpan для распространенных аллелей (фиг. 36A); тем не менее, было показано, что наблюдаемые связывающиеся пептиды, которые плохо прогнозировались посредством NetMHCIIpan, имеют очень высокую измеренную аффинность (фиг. 36B), что указывает на то, что эти наблюдения маловероятно являются ложноположительными. Примечательно, что мотивы MAPTAC™ всегда были стабильными среди множества клеточных линий (фиг. 36C).
[0755] Как правило, MAPTAC™ и IEDB согласовывались в отношении аминокислот с высокой частотой в якорных положениях (~4 наиболее высококонсервативных положения), однако мотивы MAPTAC™, главным образом, продемонстрировали более низкую энтропию (проявляющуюся более высокой высотой букв в логотипах последовательностей). Интересно, что, когда клетки сотрансфицировали конструкциями MAPTAC™ и HLA-DM, энтропия в якорных положениях еще более снижалась для большинства аллелей (фиг. 30A и фиг. 37A). Это неизменно наблюдалось для 12 аллелей HLA-DR, демонстрируя доминирующий эффект HLA-DM в качестве "редактора" репертуара и указывая на то, что модели на основе анализов аффинности, которые лишены HLA-DM и других нагружающих шаперонов, могут обучаться правилам связывания, которые не применимы in vivo. Эффект HLA-DM также был очевидным в отношении присутствия пептидов CLIP. Без сотрансфекции HLA-DM происходящие из CD74 пептиды наблюдались в 10 аллелях HLA-DR и соответствовали известным вариантам CLIP (фиг. 37C); между тем, пептиды CLIP не наблюдались ни в одном из экспериментов авторов настоящего изобретения по сотрансфекции с HLA-DM.
[0756] Эффект HLA-DM не был очевидным для проанализированных аллелей HLA-DP (фиг. 30A и фиг. 37A), что может быть связано с наличием необычного положительно заряженного якоря P1, который ранее не описан. Полагают, что HLA-DM в основном действует на N-концевой стороне связанных пептидов, сам по себе необычный якорь P1 не был маркером нечувствительности к HLA-DM, поскольку мотивы HLA-DP с гидрофобными якорями P1 также не изменялись в результате присутствия HLA-DM (фиг. 37A). С другой стороны, на HLA-DM оказывал на HLA-DQB1*06:04/A1*01:02 выраженное действие (фиг. 30A). Без сотрансфекции HLA-DM этот связывающий мотив аллеля был неопределим, что указывает на то, что свободная от шаперона загрузка на некоторые аллели HLA-DQ обеспечивает большую долю нефизиологических связывающих структур.
[0757] Учитывая доступность опубликованных мультиаллельных наборов данных для HLA класса II, проводили исследование того, могут ли аллелеспецифические пептиды быть эффективно идентифицированы с использованием способов обратной свертки in silico. Несколько групп показали успех обратной свертки мотивов аллелей HLA класса I из мультиаллельных данных для HLA класса I; однако обратная свертка мотивов HLA класса II осложнена необходимостью одновременно определять как связывающий центр, так и аллельную принадлежность каждого пептида. Для оценки точности обратной свертки для HLA класса II лигандомы HLA-DR анализировали из восьми образцов, подвергнутых определению профиля общим антителом против DR (PBMC и опубликованные клеточные линии №. Для каждого набора данных двадцать пептидов вносили в моноаллельные данные, соответствующие каждому аллелю в генотипе образца (1-2 аллеля DR1 плюс 0-2 аллеля DR3/4/5, в зависимости от гаплотипа и зиготности. Инструмент GibbsCluster (который также может использоваться для обратной свертки); использовали для распределения пептидов на группы и определения того, происходила ли соответствующая сокластеризация внесенных пептидов в соответствии с их известным аллелем происхождения. Во всех случаях пептиды распределялись в различные кластеры, демонстрируя только умеренную ассоциацию с правильными аллелями происхождения (фиг. 30B) и указывая на то, что обучающие данные HLA класса II на основе обратной свертки, вероятно, имеют значительные ошибки.
[0758] для понимания плохой эффетивности обратной свертки, рассматривали моноаллельные данные MAPTAC™ для определения частоты "очевидных" якорей, которые могут служить в качестве ориентиров для GibbsCluster. Таким образом, определяли очевидные аминокислоты (аминокислоты с частотой >10%) в каждом якорном положении (четыре положения с наиболее низкой энтропией) для каждого аллеля HLA класса II. Только 10-20% пептидов демонстрируют идеальные остатки во всех четырех якорных положениях и вплоть до 50% демонстрируют два или менее очевидных якоря (фиг. 30C). Учитывая низкую частоту пептидов, которые демонстрируют большинство из ожидаемых якорей, было неудивительно, что большую часть пептидов было бы трудно классифицировать только на вычислительной основе. Таким образом, MAPTAC™ устраняет основной источник неопределенности, что является нетривальной задачей при использовании способов in silico.
[0759] Мотивы для аллелей HLA класса I также можно было определить с использованием MAPTAC™. Это включало аллели, чьи профили связывания были ранее неопределенными (например, B*52:01, распространенный в Японии). Для ранее охарактеризованных аллелей наблюдалось, что существует хорошее соответствие между мотивами, установленными посредством способов на основе аффинности и предшествующих моноаллельных исследований посредством MS. Тем не менее было отмечено, что некоторые отличия существуют в отношении мультиаллельных исследований на основе MS, в которых использовались способы обратной свертки для определения мотивов (фиг. 37B).
Алгоритмы, обученные на данных MAPTAC™, прогнозируют иммуногенность
[0760] Рассматривали, могут ли данные MAPTAC™ обеспечивать средства прогноза связывания HLA класса II. Поскольку связывающая подпоследовательность HLA в пептидах HLA класса II не находится в фиксированном положении в отношении N- или C-конца, обучающий алгоритм должен динамически рассматривать различные возможности связывающих центров для каждого пептида. Для устранения этого ограничения использовали сверточные нейронные сети (CNN), которые являются успешными в области компьютерного зрения вследствие их умения к инвариантному к преобразованиям распознаванию паттернов. Для каждого аллеля систему CNN обучали (фиг. 31A), назвав итоговое средство прогноза "neonmhc2."
[0761] Для учета того факта, что MS демонстрирует некоторую степень смещения аминокислотных остатков, в частности, против C, получали негативные обучающие примеры (названные ловушками) посредством случайной перестановки последовательностей наблюдаемых связывающих структур (называемых наиболее подходящими последовательностями). Поскольку этот подход несет в себе риск выучивания свойств последовательностей натуральных белков, ловушки отбирали случайным образом из не выявленных подпоследовательностей генов-источников пептидов для лигандов HLA класса II. Для вычисления положительную прогностическую величину (PPV) для каждого аллеля n выявленных посредством MS пептидов оценивали совместно с 19n совпадающих по длине ловушек, взятых из того же набора генов-источников, и n пептидов с наиболее высоким рангом каждого средства прогнозирования (т.е. наивысшие 5%) называли положительными результатами. PPV в этом случае идентична отзыву, поскольку количество ложноположительных результатов и количество ложноотрицательных результатов является равным. Вычисление положительную прогностическую величину (PPV) при соотношении наиболее подходящего пептида и ловушки 1:19 показало, что PPV для neonmhc2 была лучшей относительно NetMHCIIpan в отношении прогнозирования выявленных посредством MAPTAC™ пептидов (фиг. 31B; таблица 11).
Таблица 11
[0762] Эксперименты по насыщению, в которых проводилось уменьшение набора обучающей выборки в различной степени, указывают на то, что эффективность neonmhc2 ограничена данными и вероятно повысится при увеличении количества данных (фиг. 38Ai).
[0763] Анализ, в котором наблюдалась низкая точность обратной свертки для HLA класса II на фиг. 30B, указывает на то, что сравнительная эффективность прогнозирования не может быть достигнута без моноаллельных данных. Для тестирования этого проводили недавно опубликованный вычислительный процесс, в котором используется обратная свертка для обучения средств прогноза аллелеспецифического связывания на мультиаллельных данных MS (Barra et al., 2018). При исследовании логотипов GibbsCluster для одиннадцати мультиаллельных образцов (те же образцы, что и на фиг. 30B), было обнаружено, что множество кластеров (13/32) не имели никакого сходства с известным аллелем, о котором было известно, что он находится в образце (фиг. 38Aii). С использованием существовавшего знания о том, как мотивы должны выглядеть, были отобраны только правильные кластеры (обозначенные на фиг. 38Aii) и сконструированы средства прогноза с использованием той же архитектуры CNN. Затем эти модели оценивались вместе с neonmhc2 на правильным моноаллельных данных (удержанная часть данных MAPTAC™, не использованная для обучения). Эти модели, обученные на подвергнутых обратной свертке мультиаллельных данных обычно превосходили NetMHCIIpan, но были хуже, чем обученное посредством MAPTAC™ neonmhc2 (фиг. 31E). Превосходство моноаллельных данных сохранялось, даже когда проводили уменьшение выборки для данных MAPTAC™, так чтобы размер соответствующих обучающих наборов данных был идентичным.
[0764] Чтобы убедиться, что кажущееся улучшение прогнозирования происходит при оценке не данных, полученных не посредством MS, изучали большой набор данных для аллелеспецифических ответов CD4+ T-клеток памяти, которые были обнаружены посредством тетрамер-направляемого картирования эпитопов (TGEM). Следует отметить, что эти данные для тетрамеров основаны на обмене шаперон-свободный пептид, так что он могут подвергаться тому же смещению, что и общепринятые анализы аффинности (Archila and Kwok, 2017). Тем не менее, neonmhc2 превосходил NetMHCIIpan для всех аллелей с достаточными данными для оценки (по меньшей мере 20 положительных примеров) (фиг. 31C). Эффективность (измеренная посредством PPV) NetMHCIIpan была переменной, снижаясь до 5% для DRB1*15:01 (напротив, эффективность neonmhc2 никогда не падала ниже 30% PPV), и приближалась к эффективности neonmhc2 только на двух аллелях, включая хорошо исследованный HLA-DRB1*01:01. С другой стороны, neonmhc2 продемонстрировало убедительное улучшение для всех других оцененных аллелей, включая два наиболее распространенных у европиоидов аллеля HLA-DR (DRB1*07:01 и DRB1*15:01). Эти результаты указывают на то, что усовершенствование прогнозирования neonmhc2 относительно NetMHCIIpan может быть валидировано в базе не на основе MS и, вероятно, распространяется на большинство аллелей.
[0765] Для оценки терапевтической значимости neonmhc2, проводили определение того, могло ли neonmhc2 идентифицировать неоантигены, способные индуцировать ответы CD4+ T-клеток в анализе индукции ex vivo (см. способы). Фокусируясь на DRB1*11:01, который является распространенным аллелем со многими связывающимися структурами с подтвержденной аффинностью в IEDB (его превосходят только DRB1*01:01 и DRB1*07:01; фиг. 12E), оценивали набор выявленных посредством The Cancer Genome Atlas (TCGA) последовательностей неоантигенов, и выбирали подгруппу, которая предпочиталась neonmhc2 (наивысшие 1% прогнозов), но не выбиралась NetMHCIIpan (нижние 90% прогнозов). Этот набор далее улучшали путем удаления пептидов, которые могут связывать другие аллели HLA-DR, присутствующие в подвергаемых индукции материалах. Большинство отобранных neonmhc2 пептидов (8/12) обеспечили ответы CD4+ T-клеток при измерении по экспрессии IFNγ в ответ на повторную обработку пептидом (фиг. 31D, фиг. 38B и фиг. 38C). Эти результаты демонстрируют, что обученное посредством MAPTAC™ средство прогноза может идентифицировать иммуногенные последовательности неоантигенов HLA класса II, не идентифицированные посредством NetMHCIIpan.
Профессиональные APC являются преобладающими презентирующими HLA класса II клетками в микроокружении опухоли
[0766] После разработки технологии, которая позволила как охарактеризацию, так и прогнозирование аллелеспецифических предпочтений связываний пептидов для HLA класса II, предпринималась попытка дополнить усовершенствование прогнозирования связывания дополнительной информацией о процессинге антигенов, которая является критичной для приоритезации последовательностей белков, которые наиболее вероятно будут продуцировать антигены злокачественной опухоли HLA класса II. Для решения этих вопросов в контексте TME проводили анализ данных не MAPTAC™, включающих RNA-Seq единичных клеток и опубликованные исследования на основе MS, в которых изучались лигандомы HLA класса II в опухолях. Рассматривалось, какие типы клеток в микроокружении наиболее вероятно будут презентировать злокачественные антигены для терапевтического нацеливания. В настоящее время не существует согласия в отношении того, презентируются ли злокачественные антигены профессиональными APC, поглотившими посредством эндоцитоза белки опухоли, или самими опухолевыми клетками. Для этого экспрессию HLA-DRB1 анализировали в пяти опубликованных наборах данных RNA-Seq единичных клеток для определения профиля для рака легкого, рака головы и шеи, рака ободочной и прямой кишки, рака яичника и меланомы, и обнаружили, что канонические APC (макрофаги, дендритные клетки и B-клетки) экспрессируют значительно более высокие уровни HLA класса II, чем опухолевые клетки и другие стромальные типы клеток в TME. Это наблюдение является согласующимися среди множества пациентов и типов опухолей (фиг. 19A). Поскольку опухолевые клетки могут численно превосходить APC в TME, их более низкие уровни экспрессии HLA класса II, тем не менее, могут быть иммунологически значимыми. Для оценки какая часть общей экспрессии HLA класса II происходит из опухолевых клеток против стромы, идентифицировали пациентов TCGA с мутациями в генах, специфических для HLA класса II (фокусируясь на CIITA, CD74 и CTSS) и определяли, какая часть чтений RNA-Seq демонстрировала соматический вариант, для вычисления того, какая часть экспрессии HLA-DRB1 происходила из опухоли против стромы (фиг. 19B, см. способы). На основе мутаций, идентифицированных у 153 пациентов, соответствующих 17 различным типам опухолей, оказалось, что основная часть экспрессии HLA класса II происходит в неопухолевых клетках. В действительности, 45% процентов пациентов продемонстрировали нулевую происходящую из опухоли экспрессию HLA класса II. Фокусируясь на пациентах с наиболее высокими уровнями инфильтрации T-клеток (наивысшие 10%, при идентификации с использованием ранее опубликованной сигнатуры из 18-генов (Ayers et al., 2017), низкая экспрессия HLA-DR в опухоли все еще оказывалась нормальной, причем только у 3 из 16 пациентов экспрессировалось >1000 TPM. Чтобы изучить, нарушала ли иммунотерапия эту тенденцию, авторы настоящего изобретения проанализировали дополнительные данные RNA-Seq единичных клеток для отвечающих на блокаду точек контроля типов опухолей и оценили экспрессию HLA-DRB1 до и после лечения. Группа меланомы, которая включала одного подтвержденного отвечающего пациента, продемонстрировала единообразно низкую экспрессию HLA-DRB1 опухолевыми клетками в биоптатах как до терапии, так и после терапии (фиг. 19C). Аналогично, базально-клеточная карцинома, которая продемонстрировала частоту клинического ответа 55% на терапию против PD1, аналогично продемонстрировала низкую происходящую из опухолевых клеток экспрессию HLA-DRB1 независимо от момента времени (фиг. 19C). Эти результаты указывают на то, что основная часть внутриопухолевой презентации HLA класса II определяется в основном профессиональными APC и "горячие" условия TME не гарантируют отклонения от этого общего паттерна.
Конкретные гены имеют привилегированный доступ к каскаду презентации HLA класса II
[0767] Для определения генов-источников эпитопов, которые предпочтительно презентировались находящимися в опухоли APC, и того, образуются ли они в результате аутофагии или эндоцитоза, проводили анализ трех опубликованных исследований лигандома HLA класса II, которые были проведены с использованием опухолевых тканей.
[0768] Сначала проводили количественное определение степени, с которой каждый ген был представлен в лигандомах HLA класса II опухоли, предполагая, что количество наблюдений для каждого гена должно быть пропорционально результату умножения его длины и уровня экспрессии (фиг. 18B). Наблюдалось очевидное увеличение представленности белков, экспрессируемых в плазме человека, особенно альбумина, фибриногена, факторов комплемента, аполипопротеина и трансферрина, несмотря на то, что они не экспрессировались в нативной ткани. Учитывая, что эта идентификация соответствует неспецифичскому связыванию в лигандомах HLA, проводили оценку показателей связывания neonmhc2 для происходящих из плазмы пептидов в четырех лигандомах HLA-DR PBMC (фиг. 39A); пептиды проявляли высокие показатели связывания, что указывает на то, что они были связывающими HLA. Происходящие из плазмы белки не имели представлены на значимо более высоком уровне в данных опухолевого лигандома HLA класса I (фиг. 39B). Увеличенная представленность генов плазмы в лигандомах HLA класса II согласуется с APC, "вбирающими" внеклеточные белки из сыворотки ткани посредством микропиноцитоза. Также наблюдалось дополнительное увеличение представленности для генов, вовлеченных в лейкоцитарную адгезию, таких как ITGAM (увеличение представленности в 11x), LCP1 (8x), ITGAV (6x) и ICAM1 (6x), что указывает на то, что APC активно рециклируют их собственные мембраны. MUC16, который недавно был описан как имеющий увеличенную представленность в лигандомах HLA класса I рака яичника, не имел повышенную представленность.
[0769] Клеточную локализацию также рассматривали для дальнейшего изучения генного смещения в каскаде презентации антигенов HLA класса II. Когда гены распределяли на группы по локализации, секретируемые и мембранные гены были представлены в два раза выше ожидаемого, исходя из экспрессии генов, что указывает на недооценку важной роли макропиноцитоза в формировании лигандомов HLA класса II. Тем не менее, более половины пептидов HLA класса II происходят из компартментов, не соответствующих макропиноцитозу, таких как ядро и цитоплазма. Было предположено, что, если многие из этих генов презентируются посредством аутофагии, тогда должен существовать соответствующий дефицит генов, известных тем, что выведение для них осуществляется посредством протеасомы. Действительно, белки, известные тем, что они содержат участки для убиквитина, образовывали пептиды менее часто, чем ожидалось, исходя из их длины и экспрессии (фиг. 32C). Уменьшение представленности также наблюдалось для белков, для которых известно, что их уровни возрастают при ингибировании протеасомы. Эти паттерны могут ожидаться для аутофагии, но не обязательно для фагоцитоза, что указывает на то, что лигандомы пептидов APC частично соответствуют их собственным внутриклеточным протеасомам.
[0770] Для поиска источника антигенов HLA класса II, презентируемых APC в TME, рассматривали, может ли быть возможным прямо осуществлять обратную свертку для происхождения генов-источников посредством определения, соответствовала ли идентификация пептидов в ядре и цитозоле в большей степени APC-специфическому профилю экспрессии генов или профилю экспрессии генов опухолевой массы (фиг. 39C). Хотя существовала значительная неопределенность в оценках (при оценке посредством модели на основе регрессии и бутстрэп-перевыборки; дополнительные способы), лигандомы HLA класса II наилучшим образом объяснялись смешанными профилями экспрессии как опухолью, так и APC. В совокупности с наблюдаемым уменьшением представленности выводимых протеасомами белками, этот результат указывает на то, что внутриопухолевые APC презентируют смесь экзогенных и эндогенных белков.
Некоторые области генов предпочтительно процессируются, но лишены очевидных мотивов расщепления
[0771] Существует множество теорий о том, какие последовательности являются предпочтительными для процессинга антигенов (фиг. 32D). В соответствии с одной моделью ферменты расщепляют белки-источники перед связыванием ими HLA класса II, как и в случае HLA класса I (Sercarz and Maverakis, 2003). Вторая модель предполагает, что сначала происходит связывание пептидов, а затем связанные пептиды укорачиваются экзопептидазными ферментами до тех пор, пока не сталкиваются с пространственным препятствованием посредством HLA класса II. В третей модели события расщепления пептидов происходят до и после связывания HLA. Поскольку существуют конкурирующие модели того, как образуются пептиды HLA класса II, было установлено три различных принципа для прогнозирования (фиг. 32D). Первый предполагает, что эндопептидазы преобладают ("сначала расщепление"); вторая модель предполагает, что HLA класса II связывается с полноразмерными белками, которые затем укорачиваются к центру посредством экзопептидаз ("сначала связывания"); и третья модель предполагает, что ферментативное расщепление происходит до и после связывания HLA ("гибридная"). Каждая модель требовала отдельного алгоритмического подхода. В частности, алгоритм, основанный на концепции расщепления сначала, должен фокусироваться на аминокислотных мотивах по краям выявленных посредством MS лигандов); однако для алгоритма, основанного на концепции связывания сначала, лучше сначала игнорировать эти мотивы и сфокусироваться на локальных структурных свойствах белков, которые определяют доступность HLA для связывания. Алгоритм, основанный на гибридной модели, должен осуществлять поиск участков расщепления предшественников-кандидатов выше и ниже наблюдаемых пептидов HLA класса II.
[0772] Среди трех рассмотренных подходов только алгоритм расщепления сначала обеспечивал поддающееся измерению улучшение относительно исходных моделей (фиг. 32E и фиг. 40B). Однако оказалось, что этот подход выучивает признаки укорочения экзопептидазой, присутствующие в пептидах положительных примеров (например, сигнатура предпоследнего пролина (Barra et al., 2018)), поскольку он не увеличивал значение, если точные участки расщепления пептидов запроса были скрыты (STAR Methods).
[0773] Обращаясь к чисто эмпирическому подходу, области белков, наблюдаемые в опубликованных лигандомах HLA-DQ (Bergseng et al., 2015), регистрировали и использовали перекрывание для прогнозирования лигандов HLA-DR. Переменная перекрывания обеспечивала умеренное улучшение эффективности прогнозирования (повышение PPV на 3,1% в среднем относительно neonmhc2 отдельно) (фиг. 32E). Предполагая, что аллели HLA-DQ и HLA-DR имеют одинаковые условия процессинга HLA-II, но не имеют общих связывающих мотивов, этот результат указывает на то, что определенные генные области действительно являются предпочтительными для процессинга, но они не привязаны к мотивам расщепления или конформационным свойствам очевидным образом.
[0774] Группы описали положительные результаты с использованием выявленных концов наблюдаемых посредством MS пептидов для обучения алгоритмов процессинга, подхода, который предполагает модель "сначала расщепления". Однако при рассмотрении увеличенной представленности аминокислот, соседних с концами пептидов в множестве различных клеточных линиях и типах клеток (фиг. 40A), были выявлены паттерны, которые казались более согласующимися с укорочением после связывания. Они включали отсутствие соответствия с известным мотивом фермента процессинга HLA класса II, катепсина S, и увеличением представленности плохо расщепляемого P в предпоследних положениях пептидов, мотива, который может появляться, если P блокирует процессинг посредством укорачивающих ферментов. Для тестирования того, является ли предположение "расщепления сначала" правильным, модели нейронных сетей обучали на пептидных концевых частях и оценивали их двумя различными путями: i) оценки расщепляемости на конкретных N- и C-концах каждого пептида или ii) оценки наилучшего участка в диапазоне ±15 а.к. от спрогнозированного центра связывания каждого пептида (дополнительные способы). Было предположено, что оба подхода должны увеличивать прогностическую величину, если правильной является модель расщепления сначала, однако только первый подход увеличивал ее (фиг. 32E и фиг. 40B). Таким образом, нейронная сеть может отличать HLA класса II от ловушек на основе характерных признаков в лигандах (например, предпоследний P), однако это было неприменимо, когда участки расщепления изначально неизвестны - как всегда происходит в случае прогнозирования иммуногенных пептидов и первичной белковой последовательности. Это незначительное отличие имеет потенциально приведет к запутанности в данной области.
[0775] С использованием теории "связывания сначала", выявленные посредством MS пептиды и пептиды-ловушки оценивали в отношении доступности растворителя, а также в отношении внутренне нарушенных доменов. Доступные для растворителей или нарушенные домены могут быть более представлены в лигандах HLA класса II, если белковая структура определяет доступность для связывания HLA. Однако эти признаки также оказались непрогностическими (фиг. 32E). Затем рассматривали гибридную модель, в которой ферменты частично расщепляют белок перед связыванием пептида, после чего происходит дополнительное укорочение. В этой модели, участки расщепления предшественника существуют далее выше и ниже наблюдаемых концов выявленных посредством MS лигандов. Таким образом, CNN была обучена на основе расширенного белкового контекста (±30 а.к.) для детекции отдаленных сигналов, соответствующих расщеплению предшественников. Эта модель также не продемонстрировала прогностическую величину (фиг. 32E). Наконец, предпочтительные области процессинга оказалось трудно спрогнозировать на основе первичной последовательности, области белков, выявленные в опубликованных лигандомах HLA-DQ, регистрировали и использовали перекрывание для прогнозирования лигандов HLA-DR. Переменная перекрывания обеспечивала умеренное улучшение эффективности прогнозирования (повышение PPV на 3,1% в среднем относительно neonmhc2 отдельно) (фиг. 32E). Предполагая, что аллели HLA-DQ и HLA-DR имеют одинаковые условия процессинга HLA-II, но не имеют общих связывающих мотивов, этот результат указывает на то, что определенные генные области действительно являются предпочтительными для процессинга, но они не привязаны к мотивам расщепления или конформационным свойствам очевидным образом.
Интеграция правил презентации значительно усиливает прогнозирование лигандома HLA-DR
[0776] Для количественного определения того, как правило связывания суммируется с связанными с процессингом признаками, создавали модели с множеством переменных для прогнозирования лигандомов HLA-DR для клеточных линий, презентирующих HLA класса II, дендритных клеток и мононуклеарных клеток периферической крови (PBMC) здорового донора. Хотя презентируемые пептиды не являются мутантными, сценарий прогнозирует имитирует сценарий для прогнозироавния неоантигенов, в которых случайно отобранные геномные локусы должны оцениваться с точки зрения их способности продуцировать пептиды HLA класса II. С использованием соотношения наиболее подходящих последовательностей и ловушек 1:499 и взятия ловушек случайным образом из кодирующего белок экзома, проводили оценку эффективности моделей на основе neonmhc2 и NetMHCIIpan, а также моделей, которые включали дополнительный признаки процессинга, включая установленную посредством RNA-Seq экспрессию, смещение на генном уровне (согласно фиг. 32A, см. связанную с ней фиг. 39B), и перекрывание с ранее выявленными пептидами HLA-DQ. Чтобы эта модель согласовывалась с тем, каким образом отбирались приоритетные мутантные опухолевые эпитопы-мишени для лечения злокачественной опухоли, признак смещения генного уровня модифицировали для нейтрализации предпочтений для генов плазмы, которые не являются соответствующими источниками неоантигенов.
[0777] Эти интеграционные алгоритмы подтвердили значительное улучшение как прогнозирования связывания, так и прогнозирования процессинга (фиг. 21A). В частности, полная модель продемонстрировала улучшение в от 7,4× до 61× раз относительно модели с использованием только прогнозирования связывания NetMHCIIpan, в зависимости от оцениваемого набора данных. Как экспрессия, так и генное смещение обеспечивали значительный независимый вклад в точность прогнозирования. Признак перекрывания DQ внес меньший вклад, однако неизменно обеспечивал положительное улучшение. Важно, что модели на основе аффинности были только наполовину точными по сравнению с моделями на основе MAPTAC™, даже когда они обеспечивали полную пользу связанных с процессингом переменных прогнозирования.
Сравнение точности прогнозирования с использованием происходящих из опухоли пептидов HLA класса II, презентируемых профессиональными APC
[0778] После оценки точности, достигнутой авторами изобретения, в отношении прогнозирования лигандомов HLA класса II, внимание было смещено на тестирование того, можно ли спрогнозировать происходящие из опухоли лиганды, подвергаемые эндоцитозу профессиональными APC. Наблюдение, сделанное авторами изобретения, что основная часть экспрессии HLA класса II в TME происходит в профессиональных APC, указывает на то, что этот путь процессинга, вероятно, является наиболее существенным путем для презентации опухолевых антигенов. К сожалению, общепринятые лигандомы на основе MS для опухолевых тканей не идентифицируют, какие пептиды происходят из подвергнутых эндоцитозу опухолевых белков. Таким образом, был разработан эксперимент, в котором проводили определение профиля лигандомов HLA-DR дендритных клеток (DC), которым "скармливали" SILAC-меченные опухолевые клетки (фиг. 33A).
[0779] Для мечения происходящих из опухоли белков линию злокачественных клеток с дефектом HLA класса II (K562) выращивали в среде, содержавшей изотопно меченные L и K, достигая эффективности мечения более 95%. DC скармливали либо лизированные опухолевые клетки (для имитации макропиноцитоза опухолевого дебриса), либо УФ-обработанные цельные опухолевые клетки (для имитации фагоцитоза цельных клеток). Профиль связывающих HLA-DR пептидов определяли с использованием MS для идентификации пептидов, имеющих меченные тяжелой или легкой меткой аминокислоты. Эксперимент обеспечил 29 меченных тяжелой меткой пептидов, и эксперимент с цельными клетками 56 меченных тяжелой меткой пептидов для экспериментов с лизатами и УФ-излучением, соответственно (таблица 10 (данные S1B)). Пептиды, имеющие более одной L или K, продемонстрировал полное мечение во всех кроме двух случаев, что указывает на то, что меченные тяжелой меткой пептиды происходили из опухолевых клеток, но не из вновь транслированных белков DC, которые продемонстрировали бы дискордантное мечение. Как DC без обработки, так и DC, которые были собраны после инкубации в течение 10 минут с лизатом, не обеспечивали меченные тяжелой меткой пептиды.
[0780] С использованием интегрированного алгоритма прогнозирования, описанного в настоящем описании, оценивали способность прогнозировать происходящие из опухоли пептиды. В соответствии с предшествующим результатом, полученным авторами изобретения в отношении прогнозирования природных лигандомов HLA класса II, модели на основе neonmhc2 достигали значительно более высокой точности прогнозирования, чем модели на основе NetMHCIIpan (фиг. 33D).
[0781] В отличие от генной экспрессии, признаки генного смещения и перекрывания DQ не улучшали прогнозирование подвергнутых эндоцитозу антигенов, что указывает на то, что паттерны, которые были выучены из совокупных тканевых лигандомов, не были настолько же существенными для этого класса эпитопов. При анализе генов-источников меченных тяжелой меткой пептидов, были выявлены РНК-связывающие белки (RBP), ДНК-связывающие белки (DBP), белки теплового шока (HSP) и митохондриальные белки (фиг. 21D) в противоположность преобладанию секретируемых и мембранных белков, наблюдаемому в лигандомных экспериментах (фиг. 32A). Не было очевидно, отражало ли это отличающееся предпочтение процессинга. Действительно, белки-источники, главным образом, экспрессировались на высоком в K562 (срединная экспрессия 430 TPM по сравнению с 130 TPM для немеченых пептидов), что указывает на то, что предел детекции может определять обнаруженные генные предпочтения.
[0782] Для повышения ясности конструировали модели логистической регрессии для тестирования того, может ли генная локализация и функциональные категории улучшить прогнозирование пептидов относительно моделей, которые уже учитывают экспрессию генов. RBP, DBP и HSP более не были значимыми, когда учитывали связывание и экспрессию, однако митохондриальные белки оставались значимыми (p=2,6e-4: фиг. 33E). Примечательно, что паттерн увеличения представленности полностью отличался от того, что наблюдалось меченных легкой меткой пептидах.
[0783] Для определения того может ли увеличение представленности в митохондриях улучшить прогнозирование, получали данные от нового донора с целью более глубокого охвата посредством повышения исходного клеточного материала, фокусируясь только на протоколе УФ-обработки и добавляя момент времени инкубации в течение 24 часов в дополнение к моменту времени инкубации в течение ночи. Этот эксперимент обеспечивал 77 и 59 меченных тяжелой меткой пептидов для моментов времени в течение ночи и 24 часа, соответственно, и совместно идентифицирован 78 уникальных генов-источников. С использованием модели логистической регрессии, которая учитывает митохондриальное предпочтение (выученное на первоначальных данных SILAC), авторы изобретения были способны повысить PPV на суммарное приращение 8-12% относительно моделей, которые включали только связывание и экспрессию (фиг. 33G). Это улучшение было значимым (p=1,1e-9 и p=1,5e=-8, для 16 ч и 24 ч, соответственно). Эти предпочтения не могли быть выучены из совокупных лигандомов и могут использоваться для обеспечения более точного прогнозирования эпитопов.
[0784] Наличие презентации HLA класса II в TME ассоциировано с положительными исходами у пациентов, которых лечили способами иммунотерапии злокачественной опухоли. К сожалению, неточность прогнозирования лигандов HLA класса II и неопределенность в отношении того, как опухолевые антигены презентируются в TME, замедлили разработку способов терапии, которые нацелены на антигены HLA класса II. Таким образом, была разработана моноаллельная технология определения профиля MAPTAC™, как описано в настоящем описании и были всесторонне проанализированы лигандомы опухоли для определения правил процессинга лигандов HLA класса II. MAPTAC™ позволила быстрое определение профиля для 40 аллелей HLA класса II, включая 35 аллелей HLA-DRB1, которые охватывали 95% пациентов в США. Более того, neonmhc2, алгоритм прогнозирования связывания, созданный авторами изобретения, обученный на данных MAPTAC™, превосходил NetMHCIIpan в отношении прогнозирования ответов CD4+ T-клеток памяти даже для аллелей с наибольшиим количеством существующих результатов измерения аффинности, доступных для обучения NetMHCIIpan. Было выявлено, что эффективность neonmhc2 превосходила NetMHCIIpan в отношении идентификации ответов CD4+ T-клеток памяти в валидирующем наборе данных TGEM. Более того, алгоритмы, описанные в настоящем описании, также были превосходящими в отношении прогнозирования индуцируемых ex vivo ответов CD4+ T-клеток против неоантигенов, успешно идентифицируя иммуногенные неоэпитопы, которые не были бы приоретизированы посредством NetMHCIIpan. Между тем анализ данных для опухоли, полученных посредством RNA-Seq единичных клеток, показал, что наиболее значимые опухолевые антигены, вероятно, экспрессируются преимущественно инфильтрирующими APC, фагоцитирующими опухолевые клетки. Таким образом, проводили исследование того, какие гены и генные области предпочтительно презентируются в TME и создавали модели с множеством переменных, которые в точности прогнозировали лигандомы HLA-DR и происходящие из опухоли лиганды, презентируемые фагоцитирующими APC. Эти модели значительно превышали положительную прогностическую величину NetMHCIIpan.
[0785] Преимущество прямого определения профиля эндогенно процессируемых и презентируемых лигандов HLA класса II с использованием MAPTAC™ в противоположность общепринятым способам анализа связывания пептидов состоит в том, что присутствуют загружающие пептиды шапероны, такие как HLA-DM. Известно, что HLA-DM играет роль в редактировании репертуара пептидов HLA класса II для APC, что мотивировало авторов изобретения к исследованию эффектов их дифференциальной экспрессии на лигандомы HLA класса II. Когда HLA-DM сверхэкспрессировали в экспериментах HLA-DR MAPTAC™, связывающие мотивы более отчетливо определялись, чем в экспериментах без сверхэкспрессии HLA-DM. Неожиданно, HLA-DM имел выраженный эффект на HLA-DQB1*06:04/A1*01:02, демонстрируя, что выучивание точных правил связывания пептидов для некоторых аллелей HLA-DQ может потребовать присутствия этого загружающего пептиды шаперона. Напротив, два аллеля HLA-DP не продемонстрировали эффекта (Yin et al., 2015), указывая на взаимосвязь между чувствительностью HLA-DM и предпочтениями якорей P1, которые были необычными для этих двух аллелей HLA-DP. Помимо HLA-DM, платформа MAPTAC™ обеспечивает способ быстрого обучения тому, как другие ключевые шапероны и белки, вовлеченные в каскад HLA класса II, такие как CD74 или HLA-DO, могут влиять на репертуары связываемых пептидов для аллелей HLA класса II.
[0786] Что касается опухолевой биологии, наиболее прямым наблюдением авторов изобретения было то, что APC ответственны за доминантную экспрессию HLA класса II в TME для оцениваемых типов опухоли. Это указывает на то, что презентация терапевтически значимых опухолевых антигенов, вероятно, зависит от фагоцитоза апоптотических опухолевых клеток или макропиноцитоза секретируемых опухолевых белков. Хотя существуют сообщения о прямо уничтожении CD4 T-клеток, предоставленные данные указывают на то, что CD4 T-клетки, более конкретно, играют поддерживающую роль в TME, в основном распознавая опухолевые антигены, презентируемые на инфильтрирующих лейкоцитах. Таким образом, противоопухолевые эффекты CD4 T-клеток, вероятно, по большей степени опосредуются секрецией хемокинов и цитокинов, которые регулируют транспорт и активацию других иммунных клеток, включая клетки с прямой цитолитической функцией. Хотя это является более сложным с механистической точки зрения, одно из преимуществ состоит в том, что опухоль в меньшей степени контролирует, презентировались ли антигены HLA класса II, что указывает на то, что иммунное ускользание посредством мутаций с потерей функции, частый механизм, посредством которого опухоли избегают презентации HLA класса I, может не быть настолько же частым для HLA класса II. В данной области были бы полезными последующие исследования, которые тщательно определяют, какие популяции APC ответственны за презентацию подвергнутых эндоцитозу опухолевых антигенов и существуют ли способы усиления привлечения этих фагоцитарных клеток в TME. Кроме того, было бы полезным понять, каким образом различные пути гибели опухолевых клеток, такие как гипоксия, химиотерапия и лучевая терапия, приводят к различным уровням захвата опухолевых антигенов этими APC, что может приводить к оптимальным терапевтическим комбинациям с нацеленными на HLA класса II способами терапии.
[0787] Наконец, исчерпывающий анализ лигандомов HLA класса II привел к наблюдению того, что определенные гены, по-видимому, будут представлены более часто, чем прогнозируют их уровни экспрессии транскриптов. Изучение смещения на генном уровне в опухолевых клетках способствовало улучшению прогнозирования лигандомов HLA класса II в APC; однако является возможным, что некоторые из этих сигналов менее существенны для прогнозирования неоантигенов. Например, было обнаружено увеличение представленности, которое, по-видимому, связано с аутофагией и мембранным рециклированием в APC, а не с поглощением экзогенных антигенов. Интересно, что, когда "опухолевые клетки "скармливали" дендритным клеткам in vitro, идентификация генов-источников вместо этого продемонстрировала увеличение представленности РНК-связывающих белков. Возникает желание предположить, что предпочтительно презентируются РНК-связывающие белки, поскольку такой механизм может способствовать презентации эпитопов патогенов и потенциально объяснить реактивность против РНК-связывающих белков, наблюдаемую при системной красной волчанке и других аутоиммунных состояниях. В любом случае важно отметить, что применимость процесса лигандомки HLA на основе SILAC, проводимого авторами изобретения, не ограничивается опухолевыми антигенами, поскольку она также может использоваться для исследования антигенов, вовлеченных в инфекционное заболевание и аутоиммунитет.
[0788] В общем, правила процессинга и презентации для HLA класса II являются значительно более комплексными, чем для HLA класса I. По этой причине антигены, которые запускают CD4+ T-клеточные ответы, часто остаются неопределенными. Достижения авторов изобретения в отношении определения правил связывания и процессинга для HLA класса II позволит идентификацию поддающихся нацеливанию антигенов злокачественной опухоли и других связанных с заболеванием эпитопов, которые могут быть транслированы в более эффективные способы терапии.
ПРИМЕР 13: Дополнительная информация
Обобщение экспериментов и источников данных с ассоциированными с ними мета-данными
[0789] Исчерпывающий перечень наборов данных, включая данные MAPTAC™, данные рукописей не MAPTAC™ и ранее опубликованные данные. Когда это было целесообразно, предоставлялись соответствующие ассоциированные признаки, такие как генотип образца. B) Уникальные идентификации пептидов объединяли среди экспериментальных повторений MAPTAC™, доноров PBMC, клеточных линий и экспериментов с подпиткой SILAC. Примеси и полностью трипсинизированные пептиды удаляли. См. например, по меньшей мере фиг. 12E-12F, 34A-34B, среди прочих.
Внесение пептидов для анализа обратной свертки
[0790] Иллюстративный перечень 20 иллюстративных пептидов использовали на аллель в анализе с внесением. Пептиды отбирали с требованием SPI минимум 70, длиной от 12 до 20 аминокислот, и не позволяя 9-меру перекрываться с какими-либо связывающими структурами, выявленными для других аллелей DR при определении профиля посредством MAPTAC™. Кроме того, не было двух внесенных пептидов для данного аллеля, которые имели общий 9-мер. См. например, по меньшей мере фиг. 35, 36A-36C, среди прочих.
Сводные данные TGEM для выбранных аллелей
Дополнительные экспериментальные методики
[0791] Были получены результаты для тетрамеров HLA класса II для DRB1*01:01, DRB1*03:01, DRB1*04:01, DRB1*07:01, DRB1*11:01 и DRB1*15:01 для разнообразных патогенных и аллергенных пептидов и их соответствующих прогнозов NetMHCIIpan и neonmhc2. Данные были получены из статей, опубликованных Kwok и коллегами. См. фиг. 38A-38C, среди прочих.
Дополнительные способы
Аллельные частоты для HLA класса II и статистика данных афинности, связанные с фиг. 12A и 12E
[0792] Аллельные частоты были получены с ресурса bioinformatics.bethematchclinical.org/hla-resources/haplotype-frequencies/high-resolution-hla-alleles-и-haplotypes-in-the-us-population. Набор данных mhc_ligand_full.csv загружали из данных IEDB (iedb.org/database_export_v3.php) от 21 сентября 2018 года. Для валидных показателей аффинности требовалось, чтобы "способ/технология" соответствовали следующим: "клеточный MHC/конкурентный/флуоресценция", "клеточный MHC/конкурентный/радиоактивность", "клеточный MHC/прямой/флуоресценция", "очищенный MHC/конкурентный/флуоресценция", "очищенный MHC/конкурентный/радиоактивность" или "очищенный MHC/прямой/флуоресценция", и "анализируемая группа" соответствовала следующим: "константа диссоциации KD", "константа диссоциации KD (~EC50)", "константа диссоциации KD (~IC50)", "полумаксимальная эффективная концентрация (EC50)", или "полумаксимальная ингибиторная концентрация (IC50)". Измерение приписывалось группе Søren Buus (University of Copenhagen, Дания), если строка "Buus" появлялась в поле "Авторы". В том случае, если поле авторов включало строки "Sette" или "Sidney", измерение приписывалось группе Alessandro Sette (La Jolla Institute for Immunology, США). Все другие измерения обозначались как "прочие". Для нумерации пептидов с сильным связыванием, подсчитывали только пептиды с измеренной аффинностью выше 100 нМ.
Обзор протокола MAPTAC™, связанный с фиг. 2: конструирование конструкции ДНК
[0793] Последовательности генов для аллелей HLA класса I и HLA класса II идентифицировали посредством веб-страницы IPD-IMGT/HLA (ebi.ac.uk/ipd/imgt/hla) и использовали для конструирования рекомбинантных экспрессирующих конструкций. Для HLA класса I α-цепь подвергали слиянию с C-концевым линкером GSGGSGGSAGG (SEQ ID NO: 10), а затем последовательностью биотин-акцепторной пептидной (BAP) метки GLNDIFEAQKIEWHE (SEQ ID NO: 11), стоп-кодоном и варьирующим штрихкодом ДНК, и клонировали в вектор pSF Lenti (Oxford Genetics, Оксфорд, Великобритания) через участки рестрикции NcoI и XbaI. Конструкции HLA класса II (DR, DP и DQ) аналогичным образом клонировали в pSF Lenti через участки рестрикции NcoI и XbaI, и они состояли из последовательности β-цепи, слитой на C-конце с последовательностью линкер-BAP из конструкции класса I (SGGSGGSAGGGLNDIFEAQKIEWHE (SEQ ID NO: 12)), за которой следует другой короткий линкер GSG и последовательность рибосомального пропускания F2A (VKQTLNFDLLKLAGDVESNPGP (SEQ ID NO: 13)), последовательность α-цепи, HA-метка (GSYPYDVPDYA (SEQ ID NO: 14)), стоп-кодон и варьирующий штрихкод ДНК. Для всех аллелей DR бета-цепь образовывала пару с DRA*01:01. Конструкцию HLA-DM клонировали аналогично конструкциям HLA класса II за исключением того, что она была лишена последовательности BAP и HA-метки. К HLA-DM включали в подгруппу экспериментов для HLA класса II. Идентичность всех последовательностей ДНК подтверждали посредством секвенирования по методу Сэнгера.
Культивироание клеток и временная трансфекция
[0794] Клетки Expi293 (Thermo Scientific) выращивали в среде Expi293 (Thermo Scientific) c 8% CO2 при 37°C при встряхивании при 125 об/мин. Клетки Expi293 поддерживали при плотности клеток от 0,5×106/мл до 6×106/мл с регулярным пассированием раз в две недели. 30 мл суспензии клеток Expi293 использовали для временной трансфекции при плотности клеток приблизительно 3×106/мл и жизнеспособности >90%. В кратком изложении, 30 мкг ДНК (1 мкг/мл ДНК на мл клеточной суспензии) разбавляли в 1,5 мл среды Opti-MEM (Thermo Scientific) в одной пробирке, в то время как 80 мкл реагента для трансфекции ExpiFectamineTM 293 (Thermo Scientific) разбавляли во второй пробирке, содержавшей 1,5 мл Opti-MEM. Эти две пробирки инкубировали при комнатной температуре в течение пяти минут, объединяли, осторожно перемешивали и инкубировали при комнатной температуре в течение 30 минут. К клеткам Expi293 добавляли смесь ДНК и ExpiFectamine и инкубировали при 37°C, 8% CO2, относительной влажности 80%. Через 48 ч трансфицированные клетки собирали посредством четырех технических повторений в количестве 50×106 клеток на пробирку, центрифугировали, промывали один раз 1×Gibco DPBS (Thermo Scientific) и быстро замораживали в жидком азоте для масс-спектроскопического анализа. Аликвоту 1×106 клеток собирали из каждой подвергнутой трансфекции партии и анализировали посредством вестер-блоттинга с антителом против BAP (Rockland Immunochemicals Inc., Limerick, PA) или антителом против HA (Bio-Rad, Hercules, CA) для подтверждения экспрессии меченного аффинной меткой белка HLA. Эндогенный генотип HLA класса II Expi293 был определен как DRB1*15:01, DRB1*01:01, DPB1*04:02, DPA1*01:03, DQB1*06:02, DQA1*01:02 (Laboratory Corporation of America, Burlington, NC). В некоторых экспериментах аллели HLA класса II совместно трансфицировали с HLA-DM, и в этом случае концентрацию ДНК, использованную для обеих плазмид, снижали до 0,5 мкг ДНК на мл клеточной суспензии.
[0795] Клетки A375 (ATCC) выращивали в DMEM с 10% FBS и поддерживали в культурах до смыкания монослоя не более чем на 80% с регулярным пассированием. Для экспериментов с использованием масс-спектрометрии клетки A375 культивировали в 500-см2 чашке при плотности посева 18,5×106 клеток/мл в 100 мл, как вычисляют из количества клеток смыкающегося на 70% монослоя. Через 24 часа клетки трансфицировали TransIT-X2 (Mirus Bio, Madison, WI), следуя системному протоколу TransIT с поправкой на общий объем культуры. Через 48 ч клеточную среду аспирировали и клетки промывали 1X Gibco DPBS (Thermo Scientific). Для сбора клетки A375 инкубировали в течение 10 минут при 37°C с 30 мл раствора для неферментативной диссоциации клеток (Sigma-Aldrich), центрифугировали, промывали 1X DPBS и распределяли аликвотами в количестве 50×106 клеток на образец. Клетки 293T и HeLa приобретали от ATCC и культивировали при 37°C при 5% CO2 в DMEM, 10% FBS, 2 мМ L-глутамине или DMEM+10% FBS, соответственно. Обе клеточных линии трансфицировали конструкциями HLA с использованием реагента TransIT LT1 (Mirus Bio, Madison, WI) в соответствии с инструкциями изготовителя и обрабатывали через 48 ч после трансфекции, как описано для клеток A375. Из всех образцов аликвоту 1×106 клеток собирали после каждой трансфекции и анализировали посредством вестерн-блоттинга с использованием антитела против BAP (Rockland Immunochemicals Inc., Limerick, PA) или антитела против HA (Bio-Rad, Hercules, CA) для подтверждения экспрессии меченного аффинной меткой белка HLA. Клетки B721.221 получали от Fred Hutchison Cancer Center (Seattle, WA) и культивировали в RPMI-1640 plus glutamax (Thermo Fisher Scientific) с 10% инактивированной нагреванием эмбриональной телячьей сывороткой плюс 1% пенициллин/стрептомицин (обе от Thermo Fisher Scientific). Клетки культивировали два раза в неделю и выбрасывали после 25 пассирований. Клетки K562 и клетки KG-1 (ATCC, Manassas, VA) выращивали в среде IMDM (Thermo Fisher Scientific) плюс 10% инактивированная нагреванием FBS, 1% пенициллин/стрептомицин, 1% пируват натрия и 1% MEM-NEAA. Клетки культивировали два раза в неделю и выбрасывали после 25 пассирований.
[0796] Лентивирус для трансдукции клеток B721.221, KG-1 и K562 продуцировали в клетках HEK293T, выращенных до смыкания монослоя 80%. Шесть микрограмм геномного вектора psFLenti, кодирующего HLA класса I или HLA класса II (описанного в предыдущих разделах) смешивали с 5,3 мкг лентивирусного упаковывающего вектора psPAX2 и 1,81 мкг оболочечного вектора pMD.2. ДНК смешивали с Opti-MEM (Thermo Fisher Scientific) и реагентом для трансфекции, Fugene HD (Promega, Madison, WI), и смесь инкубировали при комнатной температуре в течение 15 минут. Затем смесь капельно добавляли на чашку с клетками HEK293T и инкубировали в течение 72 часов. Затем супернатант собирали и титры лентивируса тестировали с использованием Lenti-X GoStix (Takara Bio Inc., Япония). Для трансдукции клетки высевали в 12-ячеечные планшеты с плоским дном (Corning Inc., Corning, NY) и смешивали с лентивирусным супернатантом с 6 мкг/мл полибрена (Sigma-Aldrich). Клетки, смешанные с лентвирусом, центрфугировали при 32°C при 800xg в течение 90 минут. Клетки ресуспендировали в теплой среде и инкубировали в инкубаторе при 37°C при 5% CO2 в течение 72 часов. Затем проводили селекцию клеток с использованием 1 мкг/мл пуромицина в течение 2 недель. После селекции по меньшей мере 50 миллионов клеток собирали, центрифугировали, промывали один раз 1×Gibco DPBS (Thermo Scientific), и быстро замораживали в жидком азоте для масс-спектроскопического анализа.
Экспрессия и очистка белка BirA
[0797] Использовали вектор pET19, кодирующий BirA E. coli, слитый с C-концевой гексагистидиновой меткой (SEQ ID NO: 15). Химически компетентные клетки E. coli BL21 (DE3) (New England Biolabs) трансформировали экспрессирующей BirA плазмидой (вектор pET19, кодирующий BirA E. coli, слитый с C-концевым гексагестидином (SEQ ID NO: 15)), выращивали при 37°C в среде LB плюс 100 мкг/мл ампициллина до OD600 0,6-0,8 и охлаждали до 30°C перед индукцией экспрессии добавлением 0,4 мМ изопропил-β-D-тиогалактопиранозида. Выращивание клеток E. coli продолжали при 30°C в течение 4 ч. Клетки E. coli собирали центрифугированием при 8000×g в течение 30 минут при 4°C и хранили при -80°C до применения. Замороженные клеточные осадки, экспрессирующие рекомбинантный BirA, ресуспендировали в буфере IMAC (50 мМ NaH2PO4 pH 8,0, 300 мМ NaCl) с 5 мМ имидазолом, инкубировали с 1 мг/мл лизоцима в течение 20 минут на льду и лизировали посредством обработки ультразвуком. Клеточный дебрис и нерастворимые материалы удаляли центрифугированием при 16000×g в течение 30 минут при 4°C. Затем очищенный супернатант загружали на 5-мл колонку HisTrap HP с использованием хроматографической системы ÄKTA pure (GE Healthcare), промывали буфером IMAC плюс 25 мМ и 50 мМ имидазол, а затем элюировали 500 мМ имидазолом. Фракции, содержавшие BirA, объединяли и подвергали диализу против 20 мМ Tris-HCl pH 8,0 с 25 мМ NaCl и загружали в 5-мл колонку HiTrap Q HP (GE Healthcare) и элюировали посредством применения линейного градиента от 25 до 600 мМ NaCl. Фракции, содержавшие в высокой степени чистый BirA, объединяли, подвергали замене буфера в буфере для хранения (20 мМ Tris-HCl pH 8,0 100 мМ NaCl, 5% глицерин) и концентрировали приблизительно до 5-10 мг/мл, распределяли аликвотами и быстро замораживали в жидком азоте для хранения при -80°C. Концентрацию белка BirA определяли посредством УФ-спектроскопии при OD280 нм с использованием вычисленного коэффициента экстинкции ε=47,440 M-1 см-1.
Протокол вестерн-блоттинга
[0798] Образцы добавляли в буфер для образца XT Sample Buffer и восстановитель XT Reducing Agent (Bio-Rad, Hercules, CA), нагревали при 95°С в течение пяти минут, затем объем, соответствующий ~100000 клеток, наносили на 10% гели Criterion XT Bis-Tris gels (Bio-Rad) и подвергали электрофорезу при 200 В в течение 35 минут с использованием PowerPac Basic Power Supply (Bio-Rad, Hercules, CA) с подвижным буфером XT MES Running Buffer (Bio-Rad, Hercules, CA). Гели кратковременно ополаскивали водой, затем белки переносили на PVDF-мембраны в Invitrogen iBlot Transfer Stacks (Thermo Fisher Scientific) с использованием установки P3 на устройстве Invitrogen iBlot2 Gel Transfer Device (Thermo Scientific). Для мониторинга молекулярной массы использовали Precision Plus Protein All Blue Standard (Bio-Rad, Hercules, CA). Далее мембраны промывали 3 × пять минут посредством буфера Pierce TBS Tween 20 (TBST) (25 мМ Tris, 0,15 мМ NaCl, 0,05% (об./об.) Tween 20, pH 7,5), блокировали в течение 1 ч при комнатной температуре в TBST-M (TBST, содержащая 5% (масс./об.) обезжиренное сухое молоко), затем инкубировали в течение ночи при 4°C в TBST-B (TBST, содержащая 5% (масс./об.) бычий сывороточный альбумин (Sigma Aldrich)] и разведение 1:5000 как антитела кролика против бета-тубулина (каталожный номер # ab6046, Abcam), так и антитела кролика против эпитопной метки биотинлигазы (каталожный номер №100-401-B21, Rockland Immunochemicals). Затем мембраны промывали 3 × пять минут посредством TBST, инкубировали в течение 1 ч при комнатной температуре в TBST-M, содержавшей разведение 1:10000 антитела козы против IgG кролика (конъюгированное антитело H+L-пероксидаза хрена (каталожный номер №170-6515, Bio-Rad, Hercules, CA), затем промывали при комнатной температуре 3 × пять раз посредством TBST. Наконец, мембраны инкубировали с субстратом Pierce ECL Western Blotting Substrate (Thermo Fisher Scientific, Rockford, IL), проявляли с использованием устройства для визуализации ChemiDoc XRS+ Imager (Bio-Rad) и визуализировали с использованием программного обеспечения Image Lab (Bio-Rad).
Выделение меченных аффинной меткой комплексов HLA-пептид
[0799] Выделение меченного аффинной меткой комплекса HLA-пептид проводили из клеток, экспрессирующих меченные BAP аллели HLA и отрицательные контрольные клеточные линии, которые экспрессировали только эндогенные комплексы HLA-пептид без BAP-меток. Агарозную смолу в виде гранул с нейтравидином промывали три раза 1 мл холодного PBS, а затем применяли в аффинной очистке HLA-пептида. Замороженные гранулы, содержавшие 50×106 клеток, экспрессировавших BAP-меченные пептиды HLA, размораживали на льду в течение 20 минут и осторожно лизировали посредством пипетирования вручную в 1,2 мл холодного лизирующего буфера [20 мМ Tris-Cl pH 8, 100 мМ NaCl, 6 мМ MgCl2, 1,5% (об./об.) Triton X-100, 60 мМ октилглюкозид, 0,2 мМ 2-йодацетамид, 1 мМ EDTA pH 8, 1 мМ PMSF, 1X свободный от EDTA полный коктейль ингибиторов протеаз (Roche, Basel, Швейцария)]. Лизаты инкубировали с переворачиванием при 4°C в течение 15 минут с >250 единиц нуклеазой Benzonase (Sigma-Aldrich) для деградации ДНК/РНК и центрифугировали при 15000×g при 4°C в течение 20 минут для удаления клеточного дебриса и нерастворимых материалов. Осветленные супернатанты переносили в новые пробирки и BAP-меченные пептиды HLA биотинилировали посредством инкубации с переворачиванием при комнатной температуре в течение 10 минут в 1,5-мл пробирке с 0,56 мкМ биотином, 1 мМ ATP и 3 мкМ BirA. Супернатанты инкубировали с переворачиванием при 4°C в течение 30 минут с объемом, соответствующим 200 мкл взвеси высокоемкой агарозной смолы в виде гранул с нейтравидином Pierce (Thermo Scientific) для аффинного обогащения биотинилированных комплексов HLA-пептид. Наконец, связанную с HLA смолу промывали четыре раза с 1 мл холодного буфера для промывания (20 мМ Tris-Cl pH 8, 100 мМ NaCl, 60 мМ октилглюкозид, 0,2 мМ 2-йодацетамид, 1 мМ EDTA pH 8), затем промывали четыре раза 1 мл холодного 10 мМ Tris-Cl pH 8. Между промываниями связанную с HLA смолу осторожно перемешивали вручную, а затем осаждали центрифугированием при 1500×g при 4°C в течение одной минуты. Промытую связанную с HLA смолу хранили при -80°C или сразу подвергали элюированию HLA-пептидов и обессоливанию.
Выделение комплексов HLA-пептид на основе антител
[0800] Комплексы HLA класса II DR-пептид выделяли из мононуклеарных клеток периферической крови (PBMC) здорового донора. Объем, соответствующий 75 мкл смолы GammaBind Plus Sepharose resin, промывали три раза 1 мл холодного PBS, инкубировали при переворачивании с 10 мкг антитела при 4°C в течение ночи, затем промывали три раза 1 мл холодного PBS, а затем использовали в иммунопреципитации HLA-пептид. Замороженные осадки PBMC, содержавшие 50×106 клеток, размораживали на льду в течение 20 минут и осторожно лизировали посредством пипетирования в 1,2 мл холодного лизирующего буфера [20 мМ Tris-Cl pH 8, 100 мМ NaCl, 6 мМ MgCl2, 1,5% (об./об.) Triton X-100, 60 мМ октилглюкозид, 0,2 мМ 2-йодацетамид, 1 мМ EDTA pH 8, 1 мМ PMSF, 1X полный свободный от EDTA коктейль ингибиторов протеаз (Roche, Basel, Швейцария)]. Лизаты инкубировали с переворачиванием при 4°C в течение 15 минут с >250 единиц нуклеазы Benzonase (Sigma-Aldrich) для деградации ДНК/РНК и центрифугировали при 15000×g при 4°C в течение 20 минут для удаления клеточного дебриса и нерастворимых материалов. Затем супернатанты инкубировали с переворачиванием при 4°C в течение 3 часов с антителом против HLA DR (TAL 1B5, продукт № sc-53319; Santa Cruz Biotechnology, Dallas, TX), связанным со смолой GammaBind Plus Sepharose resin (GE Life Sciences) для иммунопреципитации комплексов HLA DR-пептид. Наконец, смолу со связанным HLA промывали четыре раза с 1 мл холодного буфера для промывания (20 мМ Tris-Cl pH 8, 100 мМ NaCl, 60 мМ октилглюкозид, 0,2 мМ 2-йодацетамид, 1 мМ EDTA pH 8), затем промывали четыре раза 1 мл холодного 10 мМ Tris-Cl, pH 8. Между промываниями смолу со связанным HLA осторожно перемешивали, а затем осаждали центрифугированием при 1500×g при 4°C в течение 1 минуты. Промытую смолу со связанным HLA хранили при -80°C или сразу подвергали элюированию HLA-пептида и обессоливанию.
Элюирование HLA-пептидов и обессоливание
[0801] HLA-пептиды элюировали из меченных аффинной меткой и эндогенных комплексов HLA и одновременно обессоливали с использованием твердофазной системы экстракции Sep-Pak (Waters, Milford, MA). В кратком изложении, кассеты tC18 Sep-Pak Vac 1 cc (50 мг) с размером частиц 37-55 мкм подсоединяли к 24-позиционному распределителю для экстракции (Restek,), активировали два раза 200 мкл MeOH, а затем 100 мкл 50% (об./об.) ACN/1% (об./об.) FA, затем промывали четыре раза 500 мкл 1% (об./об.) FA. Для диссоциации HLA-пептидов из меченных аффинной меткой пептидов HLA и облегчения связывания пептидов с твердой фазой tC18, 400 мкл 3% (об./об.) ACN/5% (об./об.) FA добавляли в пробирки, содержавшие агарозную смолу в виде гранул со связанным HLA. Взвесь перемешивали пипетированием, а затем переносили в кассеты Sep-Pak. Пробирки и наконечники пипеток ополаскивали 1% (об./об.) FA (2×200 мкл) и промывные воды переносили с кассеты. 100 фмоль смеси для калибровки времени удержания Peptide Retention Time Calibration (PRTC) mixture Pierce (Thermo Scientific) добавляли в кассеты в качестве контроля загрузки. Агарозную смолу в виде гранул инкубировали два раза в течение пяти минут с 200 мкл 10% (об./об.) AcOH для дальнейшей диссоциации HLA-пептидов из меченных аффинной меткой HLA-пептидов, а затем промывали четыре раза 500 мкл 1% (об./об.) FA. HLA-пептиды элюировали с tC18 в новые 1,5-мл микропробрки (Sarstedt,) посредством ступенчатого фракционирования с использованием 250 мкл 15% (об./об.) ACN/1% (об./об.) FA, а затем 2×250 мкл 30% (об./об.) ACN/1% (об./об.) FA. Растворы, использованные для активации, загрузки образца, промывания и элюирования, проходили посредством гравитации, однако для удаления оставшегося элюата из кассет использовали вакуум (< -2,5 PSI). Элюаты, содержавшие HLA-пептиды, замораживали, сушили посредством центрифугирования в вакууме, и хранили при -80°С, а затем подвергали второму процессу обессоливания. Вторичное обессоливание образцов HLA-пептид проводили с использованием StageTips собственной конструкции, упакованных с использованием двух пуансонов калибра 16 дисков для твердофазной экстракции Empore C18 (3M, St. Paul, MN), как описано ранее. StageTips активировали два раза посредством 100 мкл MeOH, а затем 50 мкл 50% (об./об.) ACN/0,1% (об./об.) FA, а затем промывали три раза посредством 100 мкл 1% (об./об.) FA. Высушенные HLA-пептиды солюбилизировали посредством добавления 200 мкл 3% (об./об.) ACN/5% (об./об.), а затем загружали на StageTips. Пробирки и наконечники пипеток ополаскивали 1% (об./об.) FA (2×100 мкл) и объем промывочной жидкости переносили на StageTips, а затем StageTips промывали пять раз посредством 100 мкл 1% (об./об.) FA. Пептиды элюировали с использованием ступенчатого градиента объемом 20 мкл 15% (об./об.) ACN/0,1% (об./об.) FA, а затем двумя 20-мкл порциями 30% (об./об.) ACN/0,1% (об./об.) FA. Загрузку образцов, промывание и элюирование проводили на настольной центрифуге при максимальной скорости 1500-3000×g. Элюаты замораживали, сушили посредством центрифугирования в вакууме и хранили при -80°С.
Секвенирование HLA-пептид посредством тандемной масс-спектрометрии
[0802] Во всех анализах nanoLC-ESI-MS/MS использовались те же условия разделения LC, которые описаны ниже. Образцы подвергали хроматографическому разделению с использованием Proxeon Easy NanoLC 1200 (Thermo Scientific, San Jose, CA), оборудованного капиллярами с внутренним диаметром 75 мкм PicoFrit (New Objective, Inc., Woburn, MA) с 10-мкм эмиттером, упакованными под давлением 1000 фунт./кв. дюйм посредством He до ~30-40 см гранулами C18 Reprosil с размером частиц 1,9 мкм /размером пор 200 Å и нагревали при 60°C в ходе разделения. Колонку уравновешивали посредством 10X объемом слоя буфера A (0,1% (об./об.) FA и 3% (об./об.) ACN), образцы загружали в 4-мкл 3% (об./об.) ACN/5% (об./об.) FA, и пептиды элюировали посредством линейного градиента 7-30% буфера B (0,1% (об./об.) FA и 80% (об./об.) ACN) в течение 82 минут, 30-90% буфера B в течение шести минут, а затем удерживали при 90% буфере B в течение 15 минут для промывания колонки. Подгруппу образцов элюировали посредством линейного градиента 6-40% буфера B в течение 84 минут, 40-60% буфера B в течение девяти минут, а затем удерживали при 90% буфера B в течение пяти минут и 50% буфера B в течение девяти минут для промывания колонки. Линейные градиенты для элюирования образцов прогоняли при скорости 200 нл/мин и получали срединную ширину пиков ~13 с.
[0803] В ходе зависимого от данных исследования элюированные пептиды загружали в масс-спектрометр Orbitrap Fusion Lumos (Thermo Scientific), оборудованный источником ионов Nanospray Flex Ion source (Thermo Scientific) при 2,2-2,5 кВ. Полное сканирование MS проводили при разрешении 60000 с m/z от 300 до 1700 (AGC Target 4e5, максимальный IT 50 мс). После каждого полного сканирования следовало время цикла 2 с или наилучшие 10 зависимых от данных сканирований MS2 при разрешении 15000, с использованием ширины выделения 1,0 m/z, энергии столкновений 34 (данные для HLA класса) и 38 (данные для HLA класса II), ACG Target 5e4 и максимального времени заполнения 250 мс. Ширину выделения 1,0 m/z использовали, поскольку пептиды HLA класса II имеют тенденцию к большей длине (срединное значение 16 аминокислот с подгруппой пептидов из >40 аминокислот), так что моноизотопный пик не всегда является наиболее высоким в кластере изотопов, и программное обеспечение для получения данных масс-спектрометра помещает наиболее высокий изотопный пик в центр окна выделения в отсутствии указанного смещения. Таким образом, окно выделения 1,0 m/z позволяет совместное выделение моноизотопного пика, даже когда он не является наиболее высоким пиком в изотопном кластере, поскольку зарядовые состояния пептидов класса II часто представляют собой +2 или выше. Было разрешено динамическое исключение с количеством повторов 1 и длительностью исключения 5 с, чтобы позволить ~3 PSM на выбранного предшественника. Изотопы исключали, в то время как зависимые сканирования на однозарядовое состояние на предшественника было выключено, поскольку идентификация HLA-пептидов основана на качестве PSM, так что множество PSM для различных зарядовых состояний далее повышают уверенность авторов настоящего изобретения в идентификации пептидов. Скрининг зарядовых состояний для получения данных для HLA класса II был разрешен вместе с моноизотопным отбором предшественников (MIPS) с использованием режима Peptide Mode для предотвращения запуска MS/MS на ионах предшественников с зарядовым состоянием 1 (только для аллелей с основными якорными остатками), >7 или без присвоения. Для сбора данных HLA класса I, отбирали ионы предшественников с зарядовым состоянием 1 (диапазон масс 800-1700 m/z) и 2-4, в то время как зарядовые состояния >4 и без присвоения были исключены.
Интерпретация данных LC-MS/MS, связанных с фиг. 29
[0804] Спектры масс интерпретировали с использованием пакета программ Spectrum Mill v6.0 pre-Release (Agilent Technologies, Santa Clara, CA). Спектры MS/MS исключали из поиска, если они не имели MH+ предшественника в диапазоне 600-2000 (класс I)/600-4000 (класс II), имели заряд предшественника > 5 (класс I)/ >7 (класс II) или имели минимум <5 обнаруженных пиков. Объединение сходных спектров с одним и тем же m/z предшественника, полученным в одном и том же хроматографическом пике, было выключено. Для спектров MS/MS проводили поиск против базы данных, которая содержала гены UCSC Genome Browser с аннотацией hg19 генома и их кодирующие белок транскрипты (63691 записей; 10917867 уникальных 9-мерных пептидов) в комбинации с 264 часто встречающимися примесями. Перед поиском в базе данных все MS/MS должны были проходить спектральный фильтр качества длиной последовательности-метки >2, например, минимум из 3 масс, разделенных массой аминокислоты внутри цепи. Было установлен минимальный показатель расщепления основной цепи (BCS) 5, и использовалась оценочная схема ESI QExactive HLAv2. Для всех спектров нативных образцов HLA-пептид, не восстановленных и алкилированных, проводили поиск с использованием отсутствия ферментной специфичности, фиксированной модификацией цистеина в качестве цистеинилирования, со следующими переменными модификациями: окисленный метионин (m), пироглутаминовая кислота (N-концевая q), карбамидометилирование (c). Для восстановленных и алкилированных образцов HLA-пептид проводили поиск с использованием отсутствия ферментной специфичности, фиксированной модификации цистеина в качестве карбамидометилирования со следующими переменными модификациями: окисленный метионин (m), пироглутаминовая кислота (N-концевой q), цистеинилирование (c). Как для нативных, так и для восстановленных и алкилированных наборов данных HLA-пептид использовали допуск по массе предшественника ±10 м.д., допуск по массе продукта ±10 м.д., и минимальную интенсивность соответствующего пика 30%. Соответствие пептидных спектров (PSM) для индивидуальных спектров автоматически обозначалось как достоверно присвоенное с использованием модуля аутовалидации Spectrum Mill для применения оценки FDR на основе мишень-ловушка в ранге PSM для установки оценочных пороговых критериев. Стратегия автоматических пороговых значений с использованием минимальной длины последовательности 7, автоматической фильтрацией массы предшественников вариабельного диапазона, и оценки и пороговых значений оценки дельта ранг 1 - ранг 2, оптимизированных для всех прогонов LC-MS/MS для аллеля HLA, обеспечивала оценку FDR PSM <1,0% для каждого зарядового состояния предшественника.
[0805] Идентифицированные пептиды, которые проходили оценку FDR PSM <1,0%, далее фильтровали в отношении примесей путем удаления всех пептидов, отнесенных к 264 распространенным белкам-примесям в эталонной базе данных, и путем удаления пептидов, идентифицированных в аффинном анализе соосаждения MAPTAC™ для отрицательного контроля. Кроме того, все идентифицированные пептиды, которые картировались на триптическом продукте расщепления in silico эталонной базы данных, удаляли, поскольку эти пептиды не могут быть исключены из триптических примесей в результате переноса образца на колонку uPLC.
[0806] Для удаления потенциальных ложноположительных идентификаций PSM из эксперимента с подпиткой DC SILAC, применяли дополнительные фильтры качества для PSM, идентифицированных с использованием способов, описанных выше. Все пептиды с FDR<1% фильтровали в отношении высококачественных PSM с использованием следующих пороговых значений: i) оцененная интенсивность пика >60% ii) показатель расщепления основной цепи ≥8 и iii) допустимая масса в м.д. ± 1 м.д. из срединных м.д., выявленных для всех идентификаций PSM в одной и той же реплике LC-MS/MS.
Моноаллельное распределение гетеродимеров HLA-DR, -DQ, DP с использованием протокола MAPTAC™
[0807] Поскольку только бета-цепь HLA класса II является меченной в протоколе MAPTAC™, стадия соосаждения осуществляет выделение комплексов пептид-MHC независимо от того, содержат ли они нокинную или эндогенную альфа-цепь. В случае HLA-DR не считается, что аллельное варьирование в альфа-цепи влияет на связывание пептидов; таким образом, относительная степень образования пар с эндогенной цепью альфа является несущественной для интерпретации данных - данные являются эффективно моноаллельными. Однако для локусов HLA-DP и HLA-DQ альфа-цепи демонстрируют важные аллельные варианты, так что присутствие как нокинной, так и эндогенной, альфа-цепи потенциально создает 1-3 отдельных специфичностей (в зависимости от того, имеет ли клеточная линия одну или две аллели альфа-цепей и соответствует ли какая-либо из них нокинному аллелю). В принципе, эта проблема может быть смягчена путем проведения протокола с нокинной альфа-цепью и без нее и идентификации набора пептидов, специфичных к эксперименту с альфа-цепью. В данном случае использовался подход с использованием клеточной линии, которая экспрессирует единичный альфа-аллель, который соответствует нокинному альфа-аллелю.
Анализ ранее опубликованных данных MS, связанный с фиг. 12A-12F, фиг. 30A-30C, фиг. 31A-31D, фиг. 21A, фиг. 39A-39B и фиг. 40A-B
[0808] Опубликованные наборы данных LC-MS/MS, которые предоставляли файлы.raw, подвергали повторной обработке с использованием пакета программ Spectrum Mill v6.0 pre-Release (Agilent Technologies, Santa Clara, CA). Наборы данных, которые были получены на устройстве Thermo Orbitrap (например Velos, QExactive, Fusion, Lumos), в которых использовалась фрагментация HCD и набор данных MS и MS/MS в Orbitrap (высокое разрешение), анализировали с использованием параметров, описанных в приведенном выше разделе "Интерпретация данных LC-MS/MS". Для наборов данных MS и MS/MS высокого разрешения, в которых использовалась фрагментация CID, те же параметры, которые описаны выше, использовались со схемой оценки ESI Orbitrap. Для наборов данных с коллекцией данных MS в орбитрэпе и коллекцией данных MS/MS в ионной ловушке, следующие те же самые параметры, которые описаны выше, также использовались со следующими отклонениями. Для данных HCD использовали схему оценки ESI QExactive HLAv2, в то время как для данных CID использовали схему оценки ESI Orbitrap. Использовали допуск по массе предшественника ±10 м.д., допуск по массе продукта ±0,5 Да. Для наборов данных MS/MS как высокого разрешения, так и низкого разрешения, соответствие пептидных спектров (PSM) для индивидуальных спектров автоматически обозначалось как достоверно присвоенное с использованием модуля аутовалидации Spectrum Mill для применения оценки FDR на основе мишень-ловушка в ранге PSM для установки оценочных пороговых критериев. Стратегия автоматических пороговых значений с использованием минимальной длины последовательности 7, автоматической фильтрацией массы предшественников вариабельного диапазона, и оценки и пороговых значений оценки дельта ранг 1 - ранг 2, оптимизированных для всех прогонов LC-MS/MS для аллеля HLA, обеспечивала оценку FDR PSM <1,0% для каждого зарядового состояния предшественника. Анализ идентификаций пептидов из ранее опубликованных данных продемонстрировал высокую долю 9-меров (>10%). Поскольку они потенциально могут представлять собой контаминирующие лиганды HLA класса I, короткие пептиды исключали (длина <12) из всех внешних наборов данных.
Картирование пептидов на генах и "вложенных множествах", связанное с фиг. 30A-30C, фиг. 31A-31D и фиг. 32A-32E
[0809] Каждый пептид относили к одному или более кодирующим белок транскриптам в аннотации генов UCSC hg19 (genome.ucsc.edu/cgi-bin/hgTables). Поскольку многие идентификации пептидов перекрывают другие и, таким образом, составляя по большей части избыточную информацию, пептиды группировали на "вложенные множества", для каждого из которых подразумевалось соответствие ~1 уникальному событию связывания. Например, все из пептидов GKAPILIATDVASRGLDV (SEQ ID NO: 16), GKAPILIATDVASRGLD (SEQ ID NO: 17) и KAPILIATDVASRGLDV (SEQ ID NO: 18) содержат консервативную последовательность KAPILIATDVASRGLD (SEQ ID NO: 19), и, возможно, все из них связывают MHC на том же уровне. Для вложения пептидов данного набора данных строили график, в котором каждый узел соответствовал уникальному пептиду, и создавали границу между любой парой пептидов, имеющих по меньшей мере один 9-мер и картируемых по меньшей мере на одном общем транскрипте. Команду "clusters" в пакете R igraph использовали для идентификации кластеров связанных узлов, и каждый кластер был идентифицирован как вложенное множество. Эта методика гарантирует, что любые два пептида, которые удовлетворяют пограничному критерию (≥1 обычного 9-мера и ≥1 обычного транскрипта) распределяются в одно то же вложенное множество. Вложенные множества использовали для получения логотипов последовательностей (логотипы получали с использованием наиболее короткого пептида в каждом вложенном множестве; фиг. 30A-30C, машинного обучения (вес значимости среди пептидов во вложенном множестве в сумме дает единицу; фиг. 31A-31D), и анализа генного смещения (каждое вложенное множество считалось как одно наблюдение, а не каждый индивидуальный пептид; фиг. 32A-32E).
Анализ аминокислотных частот, связанный с фиг. 12F
[0810] Частоты аминокислот в протеоме человека вычисляли на основе последовательностей для всех кодирующих белок генов в аннотации UCSC hg19 (выбор одного транскрипта случайным образом для генов, соответствующих множеству изоформам транскриптов), как показано на фиг. 11D. Частоты IEDB определяли посредством идентификации уникального набора пептидов по меньшей мере с одним наблюдением аффинности ≤100 нМ (исключая некоторые пептиды c гексавалентным полигистидином на их C-конце). Частоты MAPTAC™ сначала рассматривали в контексте стандартного протокола прямой фазы для пяти аллелей DRB1 (DRB1*01:01, DRB1*03:01, DRB1*09:01, и DRB1*11:01), с использованием только одного пептида (наиболее длинного) на вложенное множество. Кроме того, частоты MAPTAC™ отдельно вычисляли для основного протокола обратной фазы среди нескольких аллелей. Данные MS из внешних наборов данных анализировали независимо от потенциального источника аллеля и аналогично с использованием наиболее длинного пептида на вложенное множество.
Конструирование логотипов последовательностей для класса I, связанное с фиг. 37B
[0811] Для каждого аллеля класса I создавали логотип последовательности длиной 9 посредством определения профилей аминокислотных частот в первых пяти положениях (картирование положений логотипов 1-5) и последних четырех положениях (картирование положения логотипов 6-9) соответствующих пептидов. Таким образом, пептиды вносили вклад в логотипы последовательностей независимо от их длины. Как и в случае логотипов для класса II, высота букв пропорциональна частоте каждой аминокислоты в каждом положении, и для аминокислот с частотой ≥10% используется кодирование цветом.
Оценка эффективности обратной свертки для пептидов HLA класса II, связанная с фиг. 30B
[0812] Для оценки способности инструмента GibbsCluster (v2.0) к кластеризации мультиаллельных данных для пептидов HLA класса II по аллелю происхождения, анализировали его эффективность на восьми образцах, включая 4 образца PBMC, 1 клеточную линию меланомы (A375) и 3 ранее опубликованных лимфобластоидных клеточных линии. Для каждого аллеля DRB1/3/4/5, присутствующего в каждом (расчетном) генотипе, было внесено двадцать пептидов из моноаллельных данных MAPTAC™, полученных авторами изобретения. Внесенные пептиды ограничивались 12-20-мерами с SPI > 70, которые не имели общего 9-мера ни с одним из пептидов в данных MAPTAC™ для других аллелей HLA-DR или ни с какими из внесенных пептидов для представляющего интерес аллеля. Затем эти усиленные наборы данных предоставляли в GibbsCluster-v2.0 с использованием установок для HLA класса II по умолчанию, за исключением вынужденного предпочтения гидрофобных остатков в положении 1, как было в других случаях для обратной свертки. Для каждого образца количество кластеров в растворе уточняли вручную и устанавливали на количество, равное количеству аллелей HLA-DR, присутствующих в генотипе.
Вычисление доли пептидов с предпочтительными якорными остатками, связанное с фиг. 30C
[0813] Якорные положения определяли как четыре положения с наиболее низкой энтропией и в этих положениях "предпочтительные" аминокислоты включали все аминокислоты с частотой ≥10%. При вычислении доли пептидов с предпочтительными аминокислотами в n положениях использовали только один пептид на вложенное множество (наиболее короткий).
Спрогнозированная аффиность выявленных посредством MS пептидов, связанная с фиг. 36A
[0814] Для каждого аллеля класса II все уникальные пептиды длиной от 14 до 17 идентифицировали и оценивали в отношении потенциала связывания с использованием NetMHCIIpan. Для сравнения проводили отбор 50000 случайных совпадающих по длине пептидов из протеома человека. Распределение плотностей определяли на основе логарифмически преобразованных величин.
Измеренная аффинность для выявленных посредством MS пептидов, связанная с фиг. 36B
[0815] Пептиды отбирали для измерения аффинности, если они имели плохую спрогнозированную аффинность связывания NetMHCIIpan (>100 нМ для DRB1*01:01 или >500 нМ для DRB1*11:01) или если они имели ≤2 эвристически определенных якорей.
Установление перекрестно валидирующих частей выборки, связанное с фиг. 31A
[0816] Строили график, в котором каждый узел соответствует кодирующему белок транскрипту и присутствуют границы между всеми парами транскриптов, обладающими по меньшей мере 5 уникальными 9-мерми аминокислотного содержимого последовательности (аннотация генов UCSC hg19). Команду "clusters" в пакете R igraph (Team, 2014) (cran.r-project.org/web/packages/igraph/citation.html) использовали для идентификации кластеров связанных узлов, и каждый кластер был идентифицирован как "группа транскриптов". Таким образом, если два транскрипта имели общую границу (≥5-общих 9-меров), они гарантированно распределялись в одну и ту же группу транскриптов. Из группы транскриптов проводили случайный отбор, разделяя протеом на восемь приблизительно равных частей. Выявленные посредством MS пептиды (и не выявленные пептиды-ловушки) помещали в части в соответствии с частью их исходных транскриптов и эти части использовали для перекрестной валидации и гиперпараметрической настройки. Графический подход к распределению протеома использовали для минимизации вероятности того, что сходные пептидные последовательности появятся в ходе обучения и оценки, что может искусственно увеличить эффективность прогнозирования.
Архитектура и обучение средства прогнозирования связывания на основе CNN для класса II, neonmhc2, связанная с фиг. 31A
[0817] Негативные примеры (ловушки) получали для обучения посредством случайной перестановки последовательностей наиболее подходящих пептидов. Этот способ получения ловушек был выбран вместо выбора невыявленных областей из протеома, чтобы устранить смещение от MS, которое может приводить к основному предпочтению аминокислот. Таким образом, средство прогноза, созданное авторами изобретения, не знает, например, об относительном снижении представленности цистеина (фиг. 12F). Аналогично, это препятствует выучиванию моделью авторов изобретения смещений MS, связанных с общими свойствами пептидов, такими как общая гидрофобность. Этот способ связан с результатами, представленными на фиг. 31A.
[0818] Модели обучали для двух сценариев применения: валидация на внутренних данных MAPTAC™ (фиг. 31B) и на внешних данных (фиг. 31C, фиг. 21A и фиг. 21B). При обучении моделей для первого из них использовалась простая процедура обучения, где оптимизация весов сети выучивалась с использованием шести частей данных (обучающие части), гиперпараметрическую оптимизацию и раннюю остановку проводили с использованием седьмой части (часть настройки), и конечную валидацию проводили на восьмой части (оценочная часть) после завершения создания модели. В случае внешней валидации использовали перекрестную валидацию, конструируя систему моделей для каждой части данных, причем эта часть удерживалась для гиперпараметрической настройки и ранней остановки, и остальные семь частей использовались для оптимизации весов сети. Кроме того, при оценке данных не MS (фиг. 31C и фиг. 31D), оценивали каждое вычитание 12-20 мера из пептида-мишени и наивысшую оценку сохраняли.
[0819] При обучении моделей авторов изобретения, вес каждой наиболее подходящей последовательности и ловушки уменьшался по функции потерь в зависимости от размера его исходного вложенного множества, так что каждое вложенное множество в целом содержало равный вес. При оценке модели в отношении гиперпараметрической настройки наиболее короткий пептид из каждого вложенного множества использовался в соответствующей части в качестве положительных примеров и перемешанные версии этих наиболее подходящих последовательностей использовались в качестве ловушек. Кроме того, применяли общий коэффициент взвешивания, так чтобы суммарный вес наиболее подходящих последовательностей был равен суммарному весу ловушек при обучении. Для конечной оценки модели, как показано на фиг. 31B, вновь из каждого вложенного множества в оценочной части (часть 8) отбирался наиболее короткий пептид, однако ловушки отбирались случайным образом из невыявленных подпоследовательностей генов-источников пептидов ("природные ловушки", описанные в следующем разделе). Таким образом, какие-либо смещения, выучиваемые этой моделью, чтобы просто отличить природные последовательности от перемешанных, не повышали эффективность, полученную авторами изобретения, в оценочной части.
[0820] Модели обучали с использованием оптимизатора Adam с первоначальной скоростью обучения 0,003, величиной beta_1 0,9, величиной beta_2 0,999 и отсутствием ловушек (параметры Keras по умолчанию за исключением скорости обучения) и использовали бинарную функцию потерь с перекрестной энтропией. Первоначальные веса моделей были выбраны с использованием инициализации He. После каждых 5 эпох обучения проводили измерение положительной прогностической величины (PPV, описанная в последующем разделе) на части настройки, и отслеживали максимальную величину. После каждой эпохи, если потери при обучении не снижались, скорость обучения умножали на 1/3. Аналогично, каждый раз, когда PPV измеряли на части настройки, если она не возрастала по сравнению с максимумом прогона, скорость обучения умножали на 1/3. Использовали схему ранней остановки, где, если потери при обучении не снижались для трех последовательных эпох или PPV настройки на повышалась выше максимума прогона на протяжении 3 последовательных проверок, то обучение останавливалось. При обучении модели использовали фиксированное соотношение наиболее подходящих последовательностей и ловушек 1:39 в обучающей выборке и 1:19 в части настройки.
[0821] Фичуризация: в то время как аминокислоты могут быть предоставлены посредством "прямого унитарного" кодирования, другие предпочитали кодировать аминокислоты с использованием матрицы PMBEC и матрицы BLOSUM (Henikoff and Henikoff, 1992), в которых сходные аминокислоты имеют сходные профили признаков. Для целей фичеризации пептидов, осуществленной авторами изобретения, получали уникальную матрицу на основе близости аминокислот в решенных белковых структурах. Идея этого подхода состоит в том, что типичные соседи аминокислот должны отражать ее химические свойства. Для каждой аминокислоты в каждой из ~100000 белковых структур DSSP (cdn.rcsb.org/etl/kabschSander/ss.txt.gz), определяли остаток, который был наиболее близким в 3D-пространстве, но находился на расстоянии по меньшей мере 10 аминокислот в первичной последовательности. С использованием этих данных определяли количество раз, когда ближайшим соседом аланина был аланин, количество раз, когда ближайшим соседом аланина был цистеин и т.д., с получением матрицы величин близости 20×20. Каждый элемент матрицы делили на результат умножения суммы соответствующих ему столбцов и рядов, и всю матрицу подвергали логарифмическому преобразованию. Наконец, из каждого элемента вычитали среднюю величину для всей матрицы.
[0822] Каждая аминокислота также кодировалась 11 бинарными признаками, описывающими свойства аминокислот, такие как то, являются ли они: кислотными (N, Q), алифатическими (I, L, V), ароматическими (H, F, W, Y), основными (H, K, R), заряженными (D, E, H, K, R), гидрофобными (A, C, F, H, I, K, L, M, T, V, W, Y), гидроксилированными (S, T), полярными (C, S, N, Q, T, D, E, H, K, R, Y, W), небольшими (V, P, A, G, C, S, T, N, D), очень маленькими (A, G, C, S) или содержащими серу (M, C). Два признака использовали для описания положения каждой аминокислоты, один из которых монотонно возрастал вдоль пептида и один из которых указывал на абсолютное расстояние от центра пептида, в обоих случаях в единицах положения (не физическое расстояние). Наконец, был включен единичный бинарный признак для указания на то, является ли аминокислота "отсутствующей" для этого положения, что могло происходить за пределами границ более коротких пептидов. Результатом является то, что каждая аминокислота кодируется 20 признаками близости аминокислот, 11 признаками свойств аминокислот, 2 признаками положения и 1 признаком отсутствующего элемента с всего 34 признаками. Все пептиды кодировались в качестве 20-меров, где центральные 20 аминокислот использовались для более длинных пептидов и величину отсутствующего элемента добавляли симметрично к краям пептидов короче 20 аминокислот.
[0823] Когда примеры вводят в нейронную сеть, как для обучения, так и для оценки, каждый из 34 признаков подвергают нормализации путем вычитания их среднего значения и деления на их стандартное отклонение. Среднее значение и стандартное отклонение вычисляют только на обучающем наборе и без учета положения в пептиде.
[0824] Для каждого аллеля систему сверточных нейронных сетей обучали для прогнозирования связывания. Схема модельной архитектуры представлена на фиг. 31A, на которой представлены два сверточных слоя с размером ядра 6 и 50 фильтрами в каждом. После каждого слоя применялся глобальный максимальный и средний пулинг и полученные величины вводились в конечный выходной нейрон с сигмоидальной активацией. Подразумевается, но не показано, что сразу после каждого сверточного слоя применялась активация ReLU, пакетная нормализация (Ioffe and Szegedy, 2015) и 20% пространственное исключение.
При обучении системы моделей для каждого аллеля архитектура была фиксированной, однако уровень регуляризации L2 варьировался. Использовали базовый вес регуляризации L2 0,05 для первого сверточного слоя и 0,1 для второго сверточного слоя. Для варьирования уровня регуляризации L2 эти величины умножали на 0,1, 0,5, и 1. Для каждой итерации в системе проводили обучение одной модели на уровень регуляризации и сохраняли наилучший, исходя из эффективности на части настройки.
Сопоставление эффективности прогнозирования на выявленных посредством MAPTAC™ пептидах, связанное с фиг. 7A
[0825] В некоторых иллюстративных способах оценки величины эффективности прогнозирования для данного пептида или белка, кодируемого аллелем HLA может использоваться способ, включающий "перемешанные ловушки". Перемешанные ловушки представляют собой пептиды, имеющие ту же длину пептидов и аминокислоты, что и у пептида, который известен тем, что он связывается с данным пептидом или белком HLA, например, на данных масс-спектроскопии, однако последовательность аминокислот перемешана. Для каждого отдельного пептида, который был идентифицирован посредством масс-спектрометрии использовали 19 таких перемешанных пептидных ловушек (наиболее подходящие последовательности:ловушки 1:19), как показано на фиг. 7A. Модель прогнозирования презентации тестировали и PPV определяли посредством анализа 5% пептидов с наилучшей оценкой в тестовой части и изучения того, какая часть из них были положительными. PPV, полученная таким образом, представлена на фиг. 7A и в таблице 12 ниже.
Таблица 12
Сопоставление эффективности прогнозирования для выявленных посредством MAPTAC™ пептидов, связанное с фиг. 31B
[0826] Для оценки эффективности прогнозирования для данного аллеля было необходимо определить набор пептидов, которые могли быть выявлены (поскольку они присутствуют в протеоме), но не выявлены в данных MS. Эти негативные примеры назвали "естественными ловушками" (в противоположность "перемешанным ловушкам", описанным выше). В качестве основных принципов, было решено следующее: распределение длины естественных ловушек должно соответствовать распределению длины выявленных посредством MS наиболее подходящих последовательностей, естественные ловушки не должны содержать последовательность, дублирующую другие естественные ловушки, естественные ловушки не должны перекрываться с наиболее подходящими последовательностями, и/или естественные ловушки должны происходить из генов, в которых присутствовала по меньшей мере одна наиболее подходящая последовательность.
[0827] Был создан следующий псевдокод, отражающий процесс оценки, удовлетворяющий этим принципам:
Инициализировать два пустых перечня наиболее подходящих последовательностей, Нминимальный и Нисчерпывающий
Для каждого вложенного множества S выявленных посредством MS пептидов:
Если ни один из пептидов в S не может быть картирован на транскрипте в части обучения или настройки:
Добавить наиболее короткий пептид в S к Нминимальный
Добавить все пептиды в S к Нисчерпывающий
Инициализировать пустой перечень пептидов-ловушек, D
Для каждого кодирующего белок транскрипта (наиболее длинный первый, наиболее короткий последний) в тестовой части:
Если ни один из пептидов в Нисчерпывающий не картируется на транскрипте:
Перейти к следующему транскрипту
Охватить белковую последовательность транскрипта набором перекрывающихся пептидов P, где длина пептидов случайным образом выбрана из распределения длин Нминимальный. Перекрывание составляет 8 аминокислот (Последний пептид в P, как правило, выступает за пределы конца белка).
Пока последний пептид в P все еще выступает:
Вычесть 1 аминокислоту из длины наиболее длинного пептида в P
Для каждого пептида в P:
Если он не имеет 9-мера с пептидом в Нисчерпывающий и он содержит 9-мер, не наблюдаемый ни в одном из пептидов в D:
Добавить пептид к D
В ином случае:
Отбраковать пептид
Нминимальный и D составляют набор данных для оценки
[0828] Для оценки эффективности в этом наборе все n наиболее подходящих пептидов оценивали посредством средства прогнозирования (neonmhc2 или NetMHCIIpan) и оценивали среди набора из 19n ловушек (случайным образом отобранные без замены из полного набора ловушек). Наивысшие 5% пептидов в объединенном наборе называли положительными результатами, и положительную прогностическую величину (PPV) вычисляли в качестве доли положительных результатов, которые представляли собой наиболее подходящие последовательности. Следует отметить, что, поскольку количество положительных результатов ограничивается количеством, равным количеству наиболее подходящих последовательностей, отзыв равен PPV в этом сценарии оценки. Применение постоянного соотношения 1:19 среди аллелей помогает стабилизировать значения эффективности, на которые в ином случае в высокой степени влияет количество наиболее подходящих последовательностей, выявленных для каждого аллеля. Это было сочтено приемлемым, поскольку было предположено, что количество наиболее подходящих последовательность в большей степени связано с экспериментальными условиями и количеством повторений, а не со свойствами, присущими аллелю.
Вычисление аффинности NetMHCIIpan для не 15-меров, связанное с фиг. 31A-D, фиг. 40A-B и 33A-D
[0829] В ранних анализах прогнозирование аффинности и процентное прогнозирование рангов посредством NetMHCIIpan-v3.1 для не 15-меров имело низкую эффективность при сравнительных анализах. Однако следующий подход значительно повышал эффективность: если пептид был длиннее 15 аминокислот, оценивали все составляющие 15-меры и отбирали наилучшее прогнозирование в качестве показателя для всего пептида; если пептид был короче 15 аминокислот, G добавляли на N-конец для доведения длины пептида до 15 и оценивали полученный удлиненный пептид.
Эффективность в зависимости от размера обучающего набора, связанная с фиг. 38A
[0830] Для понимания того, как эффективность в модели авторов изобретения ограничена размером их наборов данных, проводили анализ насыщения. Он вовлекал переобучение систем моделей при варьировании доли обучающих данных, использованного для понимания того, как это повлияет на эффективность удержанной части. На фиг. 38A представлена PPV оценочной части (часть 8) в зависимости от количества наиболее подходящих пептидов, использованных в обучающем наборе. Каждый набор данных демонстрирует среднюю PPV для набора из 10 моделей, причем планка указывает на стандартное отклонение.
Сопоставление эффективности прогнозирования естественных CD4+ T-клеточных ответов, связанное с фиг. 31C
[0831] Поскольку значительное большинство ответов CD4+ T-клеток, документированных в IEDB (tcell_full_v3.zip at iedb.org/database_export_v3.php), имеют неизвестную или вычислительно установленную рестрикцию для аллелей касса II, подгруппа записей была сфокусирована на том, что было подтверждено экспериментально посредством тетрамера класса II. Практически все такие записи были депонированы William Kwok Laboratory (Benaroya Research Institute, Seattle, WA), которые используют кровь иммунореактивных индивидуумов для проведения осуществляемого посредством тетрамера картирования эпитопов (TGEM) для различных патогенов и аллергенов. Поскольку негативные пептиды были опубликованы для некоторых исследований, но не для других, изучали исходные публикации для восстановления полного набора положительной и отрицательной реактивности пептидов. В некоторых случаях, в исходной публикации явно приводились негативные пептиды. В других случаях негативные пептиды устанавливались, следуя методикам совмещения, указанным способах публикаций, и подтверждая, что границы пептидов соответствовали известным положительным примерам. В этом анализе, приведенном на фиг. 31C, вирусные эпитопы не картировались из генов вирусов гриппа и риновирусов, и пептидные последовательности, содержащие эпитопы, использовались для прогнозирования структур, связывающихся с белком HLA класса II. В этом случае прогнозировали PPV для ответа CD4+ T-клеток памяти на пептиды для соответствующего белка HLA-DRB1. PPV определяли, задаваясь вопросом, какая часть из положительных связывающих структур эпитопов с наивысшим рангом была истинными наиболее подходящими последовательностями. Учитывая положительное образование пар между молекулой белка HLA класса II и пептидом, где молекула HLA присутствует у индивидуума, инфицированного соответствующим вирусом, у индивидуума будет индуцироваться CD4-ответ. Сравнение эффективности прогнозирования (PPV, иными словами, прогнозирование количества истинных наиболее подходящих последовательностей) между Neonmhc2 и общедоступным средством прогнозирования (NetMHCIIpan) представлено на фиг. 31C на каждый белок DRB1, протестированный в этом иллюстративном исследовании. Neonmhc2 превосходило NetMHCIIpan для каждого из шести протестированных аллелей.
[0832] Все 20-мерные пептиды оценивали посредством neonmhc2 и NetMHCIIpan. PPV вычисляли в качестве доли экспериментально подтвержденных положительных примеров среди n пептидов с наивысшей оценкой, где существовало всего n экспериментально подтвержденных пептидов (фиг. 31C).
Протокол индукции T-клеток и данные иммуногенности, связанные с фиг. 31D
[0833] Для получения дендритных клеток моноцитарного происхождения (mDC) CD14+ моноциты выделяли из моноцитов периферической крови HLA-DRB1*11:01+ здорового донора (PBMC) посредством магнитного разделения с использованием микрогранул с CD14 человека в соответствии с протоколом изготовителя (Miltenyi Biotec). Выделенные CD14+ подвергали дифференцировке в течение 5 суток в среде Cellgenix GMP DC, дополненной 800 Е/мл rh GM-CSF и 400 Е/мл rh IL-4 (Cellgenix). На 5 сутки mDC собирали и кратковременно обрабатывали 0,4 мкМ пептидом в течение 1 часа при 37 градусах Цельсия с последующим созреванием с использованием 10 нг/мл TNF-α, 10 нг/мл IL-1β, 10 нг/мл IL-6 (Cellgenix) и 0,5 нг/мл PGE1 (Cayman Pharma). Через сорок восемь часов mDC сокультивировали с аутологичными PBMC в соотношении 1:10 в среде, содержавшей AIMV/RPMI (ThermoFisher), 10% сыворотку человека (Sigma-Aldrich), 1% Pen/Strep (ThermoFisher) и дополненную 5 нг/мл IL7 и IL15 (Cellgenix). На 12 сутки T-клетки собирали и рестимулировали на созревших DC, кратковременно обработанных 0,4 мкМ пептидом, в течение 7 суток для двух дополнительных стимуляций, всего с 3 стимуляциями.
[0834] Индуцированные T-клетки метили уникальной двухцветной системной мечения штрихкодом, как описано ранее, и культивировали в течение ночи в соотношении 1:10 с обработанными пептидами и созревшими аутологичными mDC, происходящими из CD14+ моноцитов, как описано выше. На следующее утро клетки оценивали в отношении продуцирования IFN-γ в ответ на пептид посредством проточной цитометрии. Клетки обрабатывали Golgi Plug/Golgi Stop (BD Biosciences) в течение четырех часов при 37°C. Затем клетки окрашивали антителами против поверхностных маркеров CD19, CD16, CD14, CD3, CD4, CD8 (BD Biosciences, San Jose, CA), а также красителем Live/Dead Fixable Dead Cell stain (ThermoFisher); см. таблицу 13 ниже. Затем образцы подвергали пермеабилизации и фиксировали с использованием набора BD Cytofix/Cytoperm kit (BD Biosciences) в соответствии с протоколом изготовителя и окрашивали внутриклеточными антителами против IFN-γ (BD Biosciences). Образцы прогоняли через проточный цитометр BD Fortessa X-20 и анализировали с использованием программного обеспечения FlowJo (Treestar). Индуцированные образцы, которые положительно отвечали на пептид, представляли собой образцы, в которых индуцировалось продуцирование IFN-γ на 3% выше, чем в контроле без пептида.
Таблица 13
Анализ данных экспрессии HLA класса II в RNA-Seq единичных клеток, связанный с фиг. 19A
[0835] Данные RNA-Seq для единичных клеток получали из трех ранее опубликованных наборов данных, которые определяли профиль в образцах опухолей человека. Первое исследование включало данные для кожной меланомы. Файл "GSE72056_melanoma_single_cell_revised_v2.txt" загружали из Gene Expression Omnibus (ncbi.nlm.nih.gov/geo/; номер доступа: GSE72056). Клетки с обозначением статуса опухоли "2" обрабатывались как опухолевые клетки, и клетки, обозначенные посредством обозначения статуса опухоли "1" и обозначением типа иммунных клеток, равным "1"-"6", обрабатывались как T-клетки, B-клетки, макрофаги, клетки эндотелия, фибробласты и NK, соответственно. Все другие клетки исключали. Данные естественным образом предоставляли в единицах log2(TPM/10+1) и, таким образом, их математически конвертировали до масштаба TPM. После достижения шкалы TPM данные для каждой клетки подвергали перенормализации до суммы 1000000 на протяжении набора кодирующих белок символов генов UCSC (кодирующие белок гены, которые не появлялись в матрице экспрессии, безусловно обрабатывались как имеющие нулевую экспрессию). Наконец, наблюдения для единичных клеток, соответствующие тому же типу клеток и тому же биоптату-источнику, усредняли для достижения оценок экспрессии на уровне типов клеток пациента.
[0836] Второе исследование включало данные для опухолей головы и шеи. Файл "GSE103322_HNSCC_all_data.txt" загружали из Gene Expression Omnibus (ncbi.nlm.nih.gov/geo/; номер доступа: GSE103322). В соответствии с личной перепиской с Itay Tirosh (August 22, 2018), данные в этой таблице приведены в единицах log2(TPM/10+1); таким образом, величины математически конвертировали в единицы TPM. Как и в случае исследования меланомы, данные для каждой клетки подвергали перемасштабированию до суммы 1000000 на протяжении набора кодирующих белок символов генов UCSC, и наблюдения для единичных клеток, соответствующие одному и тому же типу клеток и одному и тому же исходному биоптату, усредняли. Данные, соответствующие биоптатам лимфатических узлов, исключали.
[0837] Третье исследование включало данные для немелкоклеточного рака легкого без лечения. Файлы "RawDataLung.таблица.rds" и "metadata.xlsx" загружали из ArrayExpress (ebi.ac.uk/arrayexpress/; номера доступа: E-MTAB-6149 и E-MTAB-6653). Данные (уже в единицах TPM), подвергали перемасштабированию до суммы 1000000 на протяжении набора кодирующих белок генов, как описано ранее. Наконец, наблюдения для единичных клеток, соответствующие одному и тому же типу клеток и одному и тому же биоптату-источнику, усредняли с получением оценки экспрессии на уровне типа клеток пациента. Для простоты типы клеток объединяли до более грубой степени детализации, чем первоначально описано в таблице 14.
Таблица 14
[0838] Четвертое исследование включало данные опухолей ободочной и прямой кишки. Файл "GSE81861_CRC_tumor_all_cells_FPKM.csv" загружали из Gene Expression Omnibus (ncbi.nlm.nih.gov/geo/; номер доступа: GSE81861). Единицы данных (уже в TPM) подвергали перемасштабированию до суммы 1000000 на протяжении набора кодирующих белок генов, как описано ранее. Наконец, наблюдения для единичных клеток, соответствующие одному и тому же типу клеток и одному и тому же биоптату-источнику, усредняли с получением оценки экспрессии на уровне типа клеток пациента. Для этого исследования клетки, обозначенные как "эпителий", предположительно представляют собой смесь опухолевых клеток и нормального эпителия.
[0839] Пятое исследование включало данные из серозных опухолей рака яичника. Данные секвенирования РНК единичных клеток для 6 эпителиальных злокачественных опухолей яичника от двух пациенток с низкозлокачественным серозным раком яичника (LG1, LG2) и 4 пациенток с высокозлокачественным серозным раком яичника (HG1, HG2F, HG3, HG4) были получены из других источников. Фильтрование по качеству, кластеризация и анализ соответствовали стадиям, описанным Shih et al., 2018. В кратком изложении, инструмент анализа Seurat использовали для кластеризации клеток, прошедших фильтрование по качеству (минимум 200 экспрессируемых генов, где каждый ген должен детектироваться по меньшей мере в 3 различных клетках; всего 2258 клеток). Эффекты клеточного цикла и количества уникальных транскриптов устанавливали посредством регрессии. Клетки кластеризовывали в соответствии с анализом главных компонентов и кластеры относили к типам клеток на основе экспрессии ими генных сигнатур из исходной публикации. TPM для гена HLA-DRB1 вычисляли из нормализованного уникального количества транскриптов кодирующих белок генов для каждого типа клеток для каждого пациента.
Уровни экспрессии HLA-DRB1 в этих четырех исследованиях нанесены на график на фиг. 19A.
Охарактеризация опухолевой против стромальной экспрессии класса II, связанная с фиг. 19B
[0840] Для определения относительного уровня экспрессии связывающего MHC класса II пептида, свойственного опухоли против стромы, были идентифицированные мутации в генах каскадов класса II у пациентов TCGA (называемые на основе ДНК), и для каждого пациента, имеющего мутацию класса II, проводили количественное определение относительной экспрессии мутантных и немутантных копий гена в соответствии с RNA-Seq. Кроме того, было предположено, что мутантные чтения происходят из опухоли, немутантные чтения происходят из стромы или аллелей дикого типа в опухоли, и опухоль сохраняет копию дикого типа с экспрессией, приблизительно равной экспрессии мутантной копии
[0841] Исходя из этого, бело определено, что для наблюдаемой доли мутантного аллеля f, доля экспрессии класса II, приписываемой опухоли, составляла приблизительно 2f и не более 100%. Три гена - CIITA, CD74 и CTSS - были отобраны в качестве центральных генов каскада класса II и оценены в отношении мутаций (не исключая синонимические мутации и мутации UTR) в TCGA (данные, загруженные с TumorPortal (tumorportal.org/): BRCA, CRC, HNSC, DLBCL, MM, LUAD; загруженные посредством пакетной загрузки TCGA (tcga-data.nci.nih.gov): CESC, LIHC, PAAD, PRAD, KIRP, TGCT, UCS; Synapse (synapse.org/#!Synapse:syn1729383): GBM, KIRC, LAML, UCEC, LUSC, OV, SKCM; или оригинальной публикации TCGA (cancergenome.nih.gov/publications): BLCA, KICH, STAD и THCA). Эти гены были отобраны на основе их известных ролей в экспрессии класса II и их высокой корреляции с HLA-DRB1 среди группы образцов 8500 GTEx. Другие гены с эквивалентной корреляцией с HLA-DRB1 (HLA-DRA1, HLA-DPA1, HLA-DQA1, HLA-DQB1 и HLA-DPB1) были исключены, поскольку их полиморфная природа делает их склонными к ложноположительным сигналам мутаций. В природе только небольшая доля пациентов имела мутацию в CIITA, CD74 или CTSS, и для некоторых типов опухолей отсутствовали доступные для анализа пациенты.
[0842] Последовательности из оригинального полноэкзомного секвенирования (WES) в формате Binary Sequence Alignment/Map (BAM) подвергали визуальной оценке (инструмент IGV) для подтверждения того, что мутация присутствовала в образце опухоли и не присутствовала в нормальном образце. Количества чтений мутантов против дикого типа получали из соответствующих данных RNA-Seq с использованием pysam. В целом, экспрессия HLA-DRB1 была определена на основе данных об экспрессии, загруженных с Genomic Data Commons (gdc.cancer.gov), которые были перенормализованы до суммы до 1000000 на протяжении набора кодирующих белок генов. Долю экспрессии HLA-DRB1, приписываемой опухоли (фиг. 19B), оценивали в качестве min(1,2f), где f представляет собой долю чтений RNA-Seq d CIITA, CD74 или CTSS, имеющих мутацию.
Идентификация генов, представленных на повышенном и сниженном уровне, связанная с фиг. 32A и фиг. 39B
[0843] Образцы анализировали из ранее опубликованных экспериментов с использованием MS, которые определяли профиль лигандомов MHC-II для рака яичника, рака ободочной и прямой кишки и меланомы. Многие образцы из набора данных для рака яичника имели доступные данные RNA-Seq; данные для этих образцов загружали из SRA (NCBI BioProject PRJNA398141) и выравнивали с транскриптомом UCSC hg19 с использованием средства выравнивания STAR. Для образцов яичника, которые не имели доступных данных RNA-Seq, экспрессию оценивали среди образцов с доступными данными RNA-Seq. Для исследований с раком ободочной и прямой кишки и меланомой отсутствовали соответствующие данные RNA-Seq для любых образцов, так что средние значения вычисляли для суррогатных образцов с использованием данных из TCGA (The Cancer Genome Atlas Network). Количественное определение на уровне транскрипта проводили с использованием количества транскриптов на миллион (TPM) при вычислении посредством RSEM версии 1.2.31. Оценки экспрессии далее обрабатывали путем суммирования для генного уровня, исключения некодирующих генов и перенормализации, так чтобы общая сумма TPM достигала 1000000 (перенормализация среди кодирующих белок генов учитывает варьирование от библиотеки к библиотеке уровня nc-РНК).
[0844] Для идентификации генов, представленных на повышенном и сниженном уровне в лигандомах HLA класса II, объединяли данные пяти предшествующих испытаний, которые определяли профиль в ткани яичника, ткани ободочной и прямой кишки и кожной меланомы, рака легкого и рака головы и шеи. Для каждого гена исходным предположением авторов настоящего изобретения было то, что он должен обеспечивать пептиды пропорционально его длине, умноженной на его уровень экспрессии. Для определения длины каждого гена подсчитывали уникальные 9-меры среди всех изоформ транскриптов. Экспрессию на генном уровне получали путем суммирования среди изоформ транскриптов. Наблюдаемое количество пептидов, картированных на каждом гене, определяли на уровне вложенного множества (например, пептиды GKAPILIATDVASRGLDV (SEQ ID NO: 16), GKAPILIATDVASRGLD (SEQ ID NO: 17) и KAPILIATDVASRGLDV (SEQ ID NO: 18) подсчитывались как одно наблюдение).
[0845] Создавали две матрицы, соответствующие ожидаемым и наблюдаемым количествам, обозначаемые как E и O, соответственно, где строки соответствуют генам и столбцы соответствуют образцам. Величины в O определяли путем подсчета пептидов на образец на уровне вложенного множества. Сначала заполняли матрицу E путем умножения длины каждого гена на его экспрессию в каждом образце; затем столбцы E подвергали перемасштабировали, чтобы суммы для столбцов E соответствовали суммам для столбцов O. Наконец, проводили анализ на генном уровне путем сравнения сумм строк E и сумм строк O (фиг. 32A). Гены отмечали в соответствии с их присутствием и концентрацией в плазме человека. Идентичный подход использовали для идентификации генов, представленных на повышенном и сниженном уровне в данных HLA класса I, с использованием данных для меланомы, рака ободочной и прямой кишки и рака яичника из того же набора исследований. Для анализа HLA класса I не применяли вложения, а подсчитывали только уникальные пептиды.
Оценка показателей связывания для генов с повышенной представленностью, связанная с фиг. 39A
[0846] Было выявлено, что многие из генов с повышенной представленностью представляли собой гены плазмы. Был получен исчерпывающий перечень сывороточных генов, и показатели связывания neonmhc2 сравнивали для связанных с HLA DR пептидов, происходящих из генов плазмы, со связанными с HLA-DR пептидами, происходящими из несывороточных генов, а также с совпадающими по длине несвязывающими (например, не выявленными в MS) пептидами, которые были представлены в иммунопептидоме. Для генотипированных мультиаллельных наборов данных, которые включали пептиды HLA класса II, подвергнутые определению профиля с использованием общего антитела против DR (те же образцы, что были проанализированы на фиг. 30B), оценивали пептиды с neonmhc2 для каждого аллеля DR, который экспрессировался в образце. Выход в виде наилучшего результата neonmhc2 относительно всех экспрессированных аллелей принимали в качестве репрезентативного показателя для каждого пептида. Данные объединяли для всех применимых наборов данных, и распределения показателей для каждой категории пептидов визуализировали с использованием ящичковой диаграммы.
Анализ генов, связанных с кругооборотом белков, связанный с фиг. 32C
[0847] Было идентифицировано два набора генов, предположительно отражающих белки, чей кругооборот регулировался протеасомой. Первый набор генов включал гены по меньшей мере с одним выявленным участком убиквитинилирования в клеточных линиях KG1, Jurkat или MM1S. Второй набор включал гены, чьи уровни возрастали при применении ингибитора протеасом бортезомиба (BTZ) из опубликованной статьи, применении фильтра значения p 0,01 и выборе 300 генов с наибольшим кратным изменением в сторону возрастания.
Сравнение исследовательской мощности экспрессии генов в опухолевой массе против антигенпрезентирующих клеток, связанное с фиг. 39C
[0848] Получали четыре профиля экспрессии генов. Подразумевалось, что первый соответствует APC, и его оценку проводили посредством усреднения специфических для типов клеток профилей из описанных выше экспериментов RNA-Seq единичных клеток. Усреднение включало "макрофаги" (из исследования головы и шеи, исследования легких и исследования меланомы), "CLEC9A DC" (из исследования рака легких) и "monoDC" (из исследования рака легких). Три других профиля экспрессии соответствовали профилям для опухолевой массы рака яичника, рака ободочной и прямой кишки и меланомы (данные на фиг. 19A). Профиль для яичника представлял собой среднее значение для образцов, опубликованное Schuster et al., а другие профили получены для пяти образцов TCGA с наивысшим содержанием опухолевых клеток в зависимости от типа опухоли, как ранее было установлено с использованием алгоритма "Absolute". Для каждого типа опухоли подсчитывали количество пептидов (на уровне вложенного множества) на ген и моделировали каждое количество пептидов каждого гена в качестве функции длины гена, APC-специфической экспрессии генов и опухолеспецифической экспрессии генов с использованием линейной регрессии. Выходную переменную и все входные переменные преобразовывали посредством log(x+1). С использованием модельных оценок параметров вычисляли вклад опухоли в качестве βtumor/(βtumor+βAPC), и вклад APC вычисляли в качестве βAPC/(βtumor+βAPC). Для каждого образца использовали бустрэп-перевыборку (M=100) на генном уровне для вычисления доверительных интервалов для экспериментальных соотношений.
Охарактеризация выявленных участков расщепления для пептидов HLA класса II, связанная с фиг. 40A
[0849] Естественным образом процессированные и презентированные пептиды HLA класса II анализировали из шести наборов данных: взятые PBMC, DC-подобная клеточная линия MUTZ3, ткань рака ободочной и прямой кишки, меланома, рак яичника и клеточная линия expi293. Поскольку многие пептиды имеют общий N-конец (например, GKAPILIATDVASRGLDV (SEQ ID NO: 16) и GKAPILIATDVASRGLD (SEQ ID NO: 17)) или общий C-конец (например, GKAPILIATDVASRGLD (SEQ ID NO: 17) и KAPILIATDVASRGLD (SEQ ID NO: 19)), исследовали два набора неизбыточных участка расщепления, один для N-концов и один для C-концов. Систему наименования, представленную на фиг. 41 использовали для обозначения положений выше пептидов, в пептидах и ниже пептидов. Частоты для вышележащих и нижележащих пептидов (…U1 и D1…), сравнивали против протеомных частот аминокислот и значимые отклонения оценивали посредством теста хи-квадрат. Положения пептидов (N1…C1) сравнивали против частот, наблюдаемых в пептидах MS.
Сопоставление эффективности различных средств прогнозирования расщепления для HLA класса II, связанное с фиг. 40B
[0850] Четыре образца PBMC и опубликованные наборы данных использовали для сопоставления способности связанных с расщеплением переменных/средств прогноза улучшать идентификацию презентируемых эпитопов HLA класса II.
[0851] Для создания интегрированных средств прогноза, которые прогнозируют презентацию пептидов с использованием как потенциала к связыванию, так и потенциала к расщеплению, сначала конструировали наборы данных с использованием того же подхода, который описан для фиг. 31B. Это подразумевало использование соотношения 1:19 для наиболее подходящих последовательностей и ловушек, где ловушки совпадают по длине с наиболее подходящими последовательностями и случайным образом взяты из набора генов, которые имели по меньшей мере одну наиболее подходящую последовательность. Таким образом, создавали различные наборы данных для трех различных целей:
1. Для средств прогнозирования расщепления на основе доступности растворителя и нарушения логистические модели аппроксимировали с использованием лигандомных данных для HLA класса II из опухолевых тканей человека. Было предположено, что для того, чтобы пептид был выявлен в лигандомном эксперименте, он должен быть успешно процессирован (для средств прогнозирования на основе нейронных сетей и CNN обучающие данные получали с использованием тех же наборов данных отличающимся образом, как объяснено в таблице).
2. Для оценки того, повышалась ли эффективность средства прогнозирования относительно только связывания, модели подвергали аппроксимации с использованием моноаллельных данных MAPTAC™, полученных с использованием клеток B721 и KG1, наиболее функционально сходных с APC клеточных линий. Потенциал к связыванию вычисляли с использованием neonmhc2, и логистическая регрессия определяла относительный вес, который будет присвоен переменным связывания и расщепления в прямом прогнозировании.
3. Для оцени эффективности прямого прогнозирования конструировали наборы данных для образцов PBMC и опубликованных наборов данных, аналогично тому, как описано выше. Однако, поскольку эти образцы были мультиаллельными, за показатель связывания для каждого пептида-кандидата принимали максимальную оценку для 1-4 аллелей DR, определяемых генотипом каждого донора. PPV вычисляли, как описано для фиг. 31B.
[0852] Оценивали несколько различных средств прогнозирования расщепления
Модель расщепления сначала, участок расщепления известен (нейронная сеть)
[0853] Для выучивания сигнала расщепления из выявленных посредством MS участков расщепления все уникальные 6-мерные аминокислотные последовательности от U3 до N3, и от C3 до D3 (с использованием номенклатуры, приведенной в разделе "Охарактеризация выявленных участков расщепления для пептидов HLA класса II, связанная с фиг. 40A") в лигандомах HLA класса II опухолевого происхождения, использовали в качестве положительных примеров для обучения двух различных нейронных сетей, моделирования N-концевого расщепления и C-концевого расщепления, соответственно. Как и ранее эквивалентное количество уникальных невыявленных N-концевых и C-концевых участков расщепления (отрицательные примеры) получали синтетически путем взятия аминокислотных частот в протеоме для контекста и из выявленного посредством MS лигандома для пептида. Аминокислотные последовательности кодировали посредством подгруппы тех же признаков, которые использовались в neonmhc2, в частности, близости аминокислот, исходя из белковых структур, и свойств аминокислот (например, кислотные, алифатические и т.д.). Затем для каждой из моделей N-концевых и C-концевых выявленных участков расщепления (lr=0,0005, оптимизатор Adam, бинарная перекрестная энтропия в качестве функции потерь) обучали полносвязную нейронную сеть с двумя скрытыми слоями (20 нейронов в каждом слое, а затем 10 нейронов в следующем) с активацией ReLu, с последующим конечным сигмоидальным слоем. Для регуляризации использовали частоту исключения 20%, L2-нормализацию 0,001 (только для C-концевой модели) и ограничение максимальной нормализации 4.
[0854] Для оценки пептида-кандидата применяли N-концевую модель для 6-мерной последовательности U3-N3 в отношении пептида, и применяли C-концевую модель для C3-D3. Как N-концевую, так и C-концевую, модели также применяли для 6-мерных последовательностей, совмещенных вдоль пептида-кандидата, для оценки склонности последовательности к расщеплению в самом пептиде. Обучение логистической регрессии проводили на данных MAPTAC™ с использованием показателя связывания neonmhc2, а также четырех выходных данных нейронной сети, соответствующих N-концу, C-концу и участкам расщепления с максимальной оценки для N-концевой и C-концевой моделей в пептиде.
Модель расщепления сначала, участок расщепления неизвестен (+/- а.к.) (нейронная сеть)
[0855] Для определения того, являются ли прогностическими модели расщепления, выученные на основе выявленных участков расщепления, когда конкретные концы пептидов неизвестны, те же нейронные сети, обученные, как описано выше, применяли для расширенного контекста, 15 аминокислот за пределами концов пептидов. Для оценки пептида-кандидата в этом случае вычисляли максимальную оценку для трех областей: 15 аминокислот выше пептида (независимо от положения истинного N-концевого участка расщепления), которые оценивали в N-концевой модели, пептидная последовательность, которую оценивали с использованием как N-концевой, так и C-концевой модели, и 15 аминокислот ниже пептида, которые оценивали с использованием C-концевой модели. Обучение логистической регрессии проводили на данных MAPTAC™ с использованием показателя связывания neonmhc2, а также четырех специфических для области показателей (поскольку пептид сам по себе вносит вклад в два набора величин, из N-концевой и C-концевой модели).
Модель сначала связывания, доступность растворителя
[0856] В пакете программ SCRATCH инструмент ACCpro20 использовали для прогнозирования относительной доступности для растворителя. Затем вероятность процессинга, учитывая среднюю доступность пептида для растворителя аппроксимировали к логистической регрессии с использованием данных для опухолевых тканей. Наконец, проводили обучение логистической регрессии на моноаллельных данных с использованием показателя связывания neonmhc2 и выходной информации из обученного для опухолевых тканей средства прогнозирования.
Модель связывания сначала, нарушение
[0857] Проводили оценку на остаток нарушения последовательности по всему протеому, оценивая по шкале 0-5 в соответствии с количеством двигателей прогнозирования, обозначающих положение как нарушенное (использованные серверы: anchor, espritz-d, espritz-n, espritz-x, iupred-l и iupred-s). Для каждого пептида вычисляли средний показатель нарушения, причем суммировали шесть выходных результатов средства прогнозирования нарушения. Как и в случае доступности для растворителя, первую логистическую модель аппроксимировали с использованием этого общего показателя нарушения с данными для опухолевых тканей. После этого следовало обучение логистической регрессии на моноаллельных данных с использованием показателя связывания neonmhc2 и выходных данных из средства прогнозирования, обученного для опухолевых тканей.
Гибридная модель, сканирование расщепления предшественника (+/- 30 а.к.) (CNN)
[0858] Обучающие данные для наиболее подходящих последовательностей получали, как описано для средства прогнозирования расщепления по принципу "расщепление сначала, участок расщепления известен" за исключением того, что вместо использования уникальных 6-мерных последовательностей с U3 по N3, и с C3 по D3, в качестве входных данных было взято 30 аминокислот, фланкирующих пептиды (с U30 по U1, и с D1 по D30). Более того, не проводили различение того, происходит ли 30-мерная последовательность из N-концевой или C-концевой части, а вместо этого данные объединяли для обучения единой модели для выучивания сигнала расщепления предшественника, которое предположительно могло происходить с любой стороны выявленного пептида. В этих условиях вместо использования синтетических ловушек использовали фланкирующие последовательности из невыявленных пептидов, взятые из тех же генов-источников. Последовательности кодировались, как описано выше, с использованием близости аминокислот на основе белковых структур и свойств аминокислот (например, кислотные, алифатические и т.д.). Архитектура CNN состояла из двух сверточных слоев, первый из которых имел размер ядра 2 с 48 фильтрами, за которым следовал слой с размером ядра 3 и 40 фильтрами. Эти слои имели активацию ReLu. После сверточных слоев следовал слой глобального максимального пулинга, после которого следовал конечный плотный слой с сигмоидальной активацией. Обучение CNN проводилось с частотой обучения 0,001 с оптимизацией Adam и бинарной перекрестной энтропией в качестве функции потерь.
[0859] Для оценки пептида-кандидата CNN применялась для 30 аминокислот выше и 30 аминокислот ниже пептида, генерируя показатель для N-концевой стороны и показатель для C-концевой стороны. Обучение логистической регрессии проводили на данных MAPTAC™ с использованием показателя связывания neonmhc2 и двух показателей CNN.
Перекрывание DQ
[0860] Идентификацию пептидов на основе MS объединяли среди лигандомов HLA-DQ из Bergseng et al., 2015. Создавали новый признак, отражающий то, перекрывался ли новый пептид-кандидат с одним ил этих ранее выявленных пептидов. В частности, признак принимался за 1, если он имел по меньшей мере один общий 9-мер с любым из пептидов в наборе ранее выявленных лигандов HLA-DQ; в ином случае признак принимался за 0. Обучение логистической регрессии проводилось на моноаллельных данных с использованием показателя связывания neonmhc2 и признака перекрывания.
[0861] Все из интегрированных моделей связывания и расщепления также подвергали аппроксимации и оценке с использованием NetMHCIIpan в качестве средства прогноза связывания вместо того, что на фиг. 40B.
Оценка общей эффективности прогнозирования на натуральных донорских тканях, связанная с фиг. 21A-21B
[0862] Периферическую кровь от семи здоровых доноров подвергали определению профиля с использованием DR-специфического антитела, как описано в разделе "выделение комплексов HLA-пептид на основе антител", выше. Обучающие и оценочные наборы данных конструировали с использованием алгоритма отбора наиболее подходящих последовательностей и ловушек, описанного ранее в отношении фиг. 31B. В кратком изложении, это означает репрезентацию каждого вложенного множества посредством одного наиболее подходящего пептида (наиболее короткий пептид во вложенном множестве) и наложение совпадающих по длине ловушек на гены, так чтобы они минимально перекрывались с наиболее подходящими последовательностями и минимально друг с другом. В этих условиях выбор ловушек не ограничивался выявленными посредством MS генами и вместо этого ловушки случайным образом отбирали без замены из целого протеома. Использовали соотношение наиболее подходящих последовательностей и ловушек 1:499, отражающее грубую оценку частоты презентируемых посредством HLA-DR пептидов в протеоме. Обучение логистических регрессионных моделей с показателями связывания MHC (из NetMHCIIpan или neonmhc2), а также другими входными признаками (экспрессия, генное смещение и перекрывание DQ) проводили на данных MAPTAC™ из клеточных линий KG1 и B721.
[0863] В подгруппе регрессий использовали следующие переменные, приведенные в таблице 15.
Таблица 15
[0864] Затем оценивали эффективность этих моделей на лигандомах HLA-DR из натуральной донорской ткани (образцы PBMC, и т.д.). Ловушки отбирали из протеома случайным образом (включая гены, которые никогда не продуцировали выявленный посредством MS пептид) для достижения соотношения 1:499 для наиболее подходящих последовательностей и ловушек, которое практически насыщает доступные последовательности ловушек. Для оценки (а также для обучения) использовали частоту наиболее подходящих последовательностей и ловушек 1:499. 0,2% пептидов с наивысшей оценкой в оцененном наборе данных обозначали как положительные результаты, и PPV вычисляли в качестве доли положительных клеток, которые представляли собой наиболее подходящие последовательности (см., например, фиг. 21A и таблицу 15). Следует отметить, что, поскольку количество положительных результатов ограничивается количеством, равным наиболее подходящим последовательностям, отзыв в точности равен PPV в этом оценочном сценарии. Использование неизменного соотношения 1:499 среди аллелей помогает стабилизировать уровни эффективности, на которые в ином случае в высокой степени влияет количество наиболее подходящих последовательностей, наблюдаемых для каждого донора. Это считалось приемлемым, поскольку было предположено, что количество наиболее подходящих последовательностей в большей степени связано с экспериментальными условиями, чем со свойствами, присущим донорским клеткам.
Таблица 16
Идентификация на основе SILAC презентируемых DC опухолевых пептидов, связанная с фиг. 33A
[0865] Для получения дендритных клеток моноцитарного происхождения (mDC), CD14+ моноциты выделяли из моноцитов периферической крови (PBMC) посредством магнитного разделения с использованием микрогранул с CD14 человека в соответствии с протоколом изготовителя (Miltenyi Biotec). Выделенные клетки подвергали дифференцировке в течение 6 суток в среде Cellgenix GMP DC, дополненной 800 Е/мл rh GM-CSF и 400 Е/мл rh IL-4 (Cellgenix). Клетки K562 (ATCC, Manassas, VA) подвергали изотопному мечению с использованием мечения стабильными изотопами с использованием аминокислот в клеточной культуре (SILAC). Клетки выращивали на протяжении 5 удвоений в среде RPMI 1640 для SILAC (ThermoFisher), содержавшей меченные тяжелыми изотопами аминокислоты, L-лизин 2HCl 13C6 15N2 (Life Technologies) и L-лейцин 13C6 (Life Technologies) с 15% инактивированной нагреванием подвергнутой диализу эмбриональной телячьей сывороткой (ThermoFisher). Меченные SILAC клетки K562 лизировали с использованием 60 мкМ гипохлорной кислоты (HOCl), как описано ранее, или обрабатывали посредством УФ-излучения в течение 3 часов при комнатной температуре для индукции апоптоза, и оставляли в покое на ночь. Семьдесят пять миллионов mDC сокультивировали с обработанными УФ SILAC-меченными клетками K562 в соотношении 1:3 в течение 14 часов при 37°C или культивировали в соотношении 1:3 с K562, лизированными посредством HOCl в течение 10 минут или 5 часов при 37°C. После сокультивирования клетки собирали, осаждали и быстро замораживали в жидком азоте для протеомного анализа.
Анализ прогнозирования и экспрессии презентируемых DC опухолевых пептидов, связанный с фиг. 33B и фиг. 21C
[0866] Для вычисления PPV для прогнозирования меченных тяжелой меткой (происходящих из опухоли) пептидов использовали ту же модель и подход оценки, которые использовались на фиг. 33B. Экспрессию для клеточных линий K562 определяли на основе данных из ENCODE (encodeproject.org/experiments/ENCSR545DKY/; библиотеки ENCLB075GEK и ENCLB365AUY; (ENCODE Project Consortium, 2012)). Экспрессию для дендритных клеток определяли на основе GSE116412 (усредняющая для последовательностей с номером доступа GSM3231102, GSM3231111, GSM3231121, GSM3231133, GSM3231145).
Пример 14. Сопоставление FAIMS с триптическими пептидами
[0867] В этом примере описан стандартный HLA-пептидомный процесс с использованием спектрометрии подвижности ионов в несимметричном поле высокой напряженности (FAIMS). Проводили охарактеризацию эндогенно процессированных и презентированных пептидов HLA класса I и HLA класса II из клеток A375. Пептиды подвергали как кислотному обращенно-фазовому (aRP), так и основному обращенно-фазовому (bRP) фракционированию вне системы перед анализом посредством nLC-MS/MS с использованием масс-спектрометра Thermo Scientific Orbitrap Fusion Lumos Tribrid, оборудованного (+) интерфейсом FAIMS Pro или не оборудованного им (-). Процесс указан на диаграмме, представленной на фиг. 42A. На фиг. 42B представлены результаты, указывающие на то, что FAIMS улучшает детекцию пептидов в триптических образцах до всего 10 нг. FAIMS повышает детекцию пептидов HLA-1 и HLA класса II посредством градиента LC, несмотря на более низкую интенсивность MS1 (фиг. 43A и фиг. 44A, данные для пептидов HLA-1 и HLA класса II, соответственно). Неожиданно, повышение детекции уникальных пептидов выявлено в образцах кислотного и основного обращенно-фазового исследования с оценкой FAIMS (фиг. 43B и фиг. 44B, данные для пептидов HLA-1 и HLA класса II, соответственно). Это исследование показало, что детекция пептидов HLA класса I и HLA класса II возрастает на протяжении градиента LC при использовании FAIMS, несмотря на более низкую интенсивность MS1. Фракционирование вне системы и FAIMS в совокупности повышают глубину анализа репертуаров пептидов HLA, как показано на фиг. 45 и фиг. 46 (пептиды HLA-1 и HLA класса II, соответственно).
Пример 15 - Анализ пептидного обмена с использованием дифференциальной сканирующей флуориметрии (DSF)
[0868] Анализ пептидного обмена проводили следующим образом: следующие реагенты (таблица 17) объединяли и перемешивали при 37°C в течение 18 часов.
Таблица 17
A Использовали DMSO или пептиды со следующими последовательностями: PPIDGYPNHPCFEPE (SEQ ID NO: 31) (M230), PQILPYPAPEEAQEN (SEQ ID NO: 32) (M231), PQLRQWWAQGADPLA (SEQ ID NO: 33) (M247), LLRPGQIVAFDSTAQ (SEQ ID NO: 34) (M248) или ASLRSWPSTWAPWAS (SEQ ID NO: 35) (M371.
[0869] Затем буфер подвергали обмену с использованием колонки для обессоливания PD minitrap G-25. Оранжевый краситель Sypro (Fisher S6651) разбавляли до 1000X в 100% DMSO. 50 мкл рабочего исходного раствора оранжевого красителя Sypro в концентрации 100X получали в буфере для обессоливания. 2 мкл 100X оранжевого красителя sypro и 18 мкл обессоленного образца для пептидного обмена переносили в лунки 384-луночного белого микропланшета для ПЦР и перемешивали. Затем планшет накрывали прозрачным герметизирующим материалом и планшет подвергали следующей программа в Roche lightcycler 480: (1) нагревание до 25°C, удержание в течение 10 секунд; (2) повышение температуры до 99°C, считывание планшета 20 раз/1°C (3) снижение температуры до 25°C и удержание в течение 10 секунд. Затем вычисляли температуру плавления. Иллюстративные результаты представлены в таблице 18 ниже.
Таблица 18
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> NEON THERAPEUTICS, INC.
<120> СПОСОБ И СИСТЕМЫ ДЛЯ ПРОГНОЗИРОВАНИЯ СПЕЦИФИЧЕСКИХ ДЛЯ HLA КЛАССА II
ЭПИТОПОВ И ОХАРАКТЕРИЗАЦИИ CD4+ T-КЛЕТОК
<130> 50401-735.601
<140> PCT/US2019/068084
<141> 2019-12-20
<150> 62/891,101
<151> 2019-08-23
<150> 62/855,379
<151> 2019-05-31
<150> 62/826,827
<151> 2019-03-29
<150> 62/783,914
<151> 2018-12-21
<160> 96
<170> PatentIn version 3.5
<210> 1
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 1
Pro Val Ser Lys Met Arg Met Ala Thr Pro Leu Leu Met Gln Ala
1 5 10 15
<210> 2
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 2
Pro Glu Ala Ser Leu Tyr Gly Ala Leu Ser Lys Gly Ser Gly Gly
1 5 10 15
<210> 3
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 3
Pro Ala Thr Tyr Ile Leu Ile Leu Lys Glu Phe Cys Leu Val Gly
1 5 10 15
<210> 4
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<220>
<221> ДОПОЛНИТЕЛЬНЫЙ ПРИЗНАК
<222> (1)..(15)
<223> Эта последовательность может охватывать 4-15 остатков
<400> 4
His His His His His His His His His His His His His His His
1 5 10 15
<210> 5
<211> 8
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 5
His His His His His His His His
1 5
<210> 6
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 6
Glu Gly Arg Gly Ser Leu Thr Cys Gly Asp Val Glu Asn Pro Gly Pro
1 5 10 15
<210> 7
<211> 17
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 7
Ala Thr Asn Phe Ser Leu Lys Gln Ala Gly Asp Val Glu Asn Pro Gly
1 5 10 15
Pro
<210> 8
<211> 19
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 8
Gln Cys Thr Asn Tyr Ala Leu Lys Leu Ala Gly Asp Val Glu Ser Asn
1 5 10 15
Pro Gly Pro
<210> 9
<211> 21
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 9
Val Lys Gln Thr Leu Asn Phe Asp Leu Lys Leu Ala Gly Asp Val Glu
1 5 10 15
Ser Asn Pro Gly Pro
20
<210> 10
<211> 11
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 10
Gly Ser Gly Gly Ser Gly Gly Ser Ala Gly Gly
1 5 10
<210> 11
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 11
Gly Leu Asn Asp Ile Phe Glu Ala Gln Lys Ile Glu Trp His Glu
1 5 10 15
<210> 12
<211> 25
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 12
Ser Gly Gly Ser Gly Gly Ser Ala Gly Gly Gly Leu Asn Asp Ile Phe
1 5 10 15
Glu Ala Gln Lys Ile Glu Trp His Glu
20 25
<210> 13
<211> 22
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 13
Val Lys Gln Thr Leu Asn Phe Asp Leu Leu Lys Leu Ala Gly Asp Val
1 5 10 15
Glu Ser Asn Pro Gly Pro
20
<210> 14
<211> 11
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 14
Gly Ser Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
1 5 10
<210> 15
<211> 6
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 15
His His His His His His
1 5
<210> 16
<211> 18
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 16
Gly Lys Ala Pro Ile Leu Ile Ala Thr Asp Val Ala Ser Arg Gly Leu
1 5 10 15
Asp Val
<210> 17
<211> 17
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 17
Gly Lys Ala Pro Ile Leu Ile Ala Thr Asp Val Ala Ser Arg Gly Leu
1 5 10 15
Asp
<210> 18
<211> 17
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 18
Lys Ala Pro Ile Leu Ile Ala Thr Asp Val Ala Ser Arg Gly Leu Asp
1 5 10 15
Val
<210> 19
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 19
Lys Ala Pro Ile Leu Ile Ala Thr Asp Val Ala Ser Arg Gly Leu Asp
1 5 10 15
<210> 20
<211> 10
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 20
His His His His His His His His His His
1 5 10
<210> 21
<211> 24
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 21
Leu Pro Lys Pro Pro Lys Pro Val Ser Lys Met Arg Met Ala Thr Pro
1 5 10 15
Leu Leu Met Gln Ala Leu Pro Met
20
<210> 22
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 22
Leu Pro Leu Lys Met Leu Asn Ile Pro Ser Ile Asn Val His
1 5 10
<210> 23
<211> 13
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 23
Pro Lys Tyr Val Lys Gln Asn Thr Leu Lys Leu Ala Thr
1 5 10
<210> 24
<211> 17
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 24
Pro Val Val His Phe Phe Lys Asn Ile Val Thr Pro Arg Thr Pro Pro
1 5 10 15
Tyr
<210> 25
<211> 17
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 25
Tyr Ala Thr Phe Phe Ile Lys Ala Asn Ser Lys Phe Ile Gly Ile Thr
1 5 10 15
Glu
<210> 26
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 26
Thr Leu Ser Val Thr Phe Ile Gly Ala Ala Pro Leu Ile Leu Ser Tyr
1 5 10 15
<210> 27
<211> 17
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 27
Pro Val Val His Phe Phe Lys Asn Ile Val Thr Pro Arg Thr Pro Pro
1 5 10 15
Tyr
<210> 28
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 28
Tyr Ala Arg Ile Arg Arg Asp Gly Cys Leu Leu Arg Leu Val Asp
1 5 10 15
<210> 29
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 29
Thr Leu Ser Val Thr Phe Ile Gly Ala Ala Pro Lys Ile Leu Ser Tyr
1 5 10 15
<210> 30
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 30
Tyr Ala Arg Ile Lys Arg Asp Gly Cys Leu Leu Arg Leu Val Asp
1 5 10 15
<210> 31
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 31
Pro Pro Ile Asp Gly Tyr Pro Asn His Pro Cys Phe Glu Pro Glu
1 5 10 15
<210> 32
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 32
Pro Gln Ile Leu Pro Tyr Pro Ala Pro Glu Glu Ala Gln Glu Asn
1 5 10 15
<210> 33
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 33
Pro Gln Leu Arg Gln Trp Trp Ala Gln Gly Ala Asp Pro Leu Ala
1 5 10 15
<210> 34
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 34
Leu Leu Arg Pro Gly Gln Ile Val Ala Phe Asp Ser Thr Ala Gln
1 5 10 15
<210> 35
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 35
Ala Ser Leu Arg Ser Trp Pro Ser Thr Trp Ala Pro Trp Ala Ser
1 5 10 15
<210> 36
<211> 9
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 36
Gly Lys Ser Val Val Cys Glu Ala Leu
1 5
<210> 37
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 37
Leu Pro Asn Gly Gly Phe Ala Ser Ile Leu Leu Tyr Lys Ile Glu
1 5 10 15
<210> 38
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 38
Phe Cys Lys Gly Ser Phe Ala Ser Ile Leu Lys Leu Leu Gly Glu Phe
1 5 10 15
<210> 39
<211> 42
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический
polypeptide
<220>
<221> MOD_RES
<222> (6)..(19)
<223> Any amino acid
<400> 39
Gly Asp Thr Gly Leu Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10 15
Xaa Xaa Xaa Gly Gly Gly Gly Ser Leu Val Pro Arg Gly Ser Gly Gly
20 25 30
Gly Gly Ser Gly Asp Thr Arg Pro Arg Phe
35 40
<210> 40
<211> 13
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 40
Asp Arg Tyr Glu Met Glu Asp Gly Lys Val Ile Glu Arg
1 5 10
<210> 41
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 41
Gly Gly His Met Thr Thr Leu Ser Gly Glu Glu Ile Ser Tyr Thr Gly
1 5 10 15
<210> 42
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 42
Lys Thr Phe Asp Gln Leu Thr Pro Glu Glu Ser Lys Glu Arg
1 5 10
<210> 43
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 43
Leu Pro Arg Tyr Glu Ala Leu Arg Gly Glu Gln Pro Pro Asp
1 5 10
<210> 44
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 44
Asp Lys Lys Asn Ile Ile Leu Glu Glu Gly Lys Glu Ile Leu Val Gly
1 5 10 15
<210> 45
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 45
Lys Glu Ala Ala Tyr His Pro Glu Val Ala Pro Asp Val Arg
1 5 10
<210> 46
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 46
Asp Ala Glu Phe Arg His Asp Ser Gly Tyr Glu Val His His Gln
1 5 10 15
<210> 47
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 47
Gly His Pro Asp Leu Gln Gly Gln Pro Ala Glu Glu Ile Phe Glu Ser
1 5 10 15
<210> 48
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 48
Leu Gly Lys Asn Phe Asp Phe Gln Lys Ser Asp Arg Ile Asn Ser Glu
1 5 10 15
<210> 49
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 49
Met Pro Ser Phe Val Pro Ser Asp Gly Arg Gln Ala Ala Asp
1 5 10
<210> 50
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 50
Gly Leu Arg Tyr Lys Lys Leu His Asp Pro Lys Gly Trp Ile Thr
1 5 10 15
<210> 51
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 51
Thr Ile Glu Lys Phe Glu Lys Glu Ala Ala Glu Met Gly Lys Gly
1 5 10 15
<210> 52
<211> 13
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 52
Gly Val Gln Arg Gly Leu Val Gly Glu Ile Ile Lys Arg
1 5 10
<210> 53
<211> 11
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 53
Thr Trp Phe Asn Gln Pro Ala Arg Lys Ile Arg
1 5 10
<210> 54
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 54
Glu Phe Lys Lys Tyr Glu Met Met Lys Glu His Glu Arg Arg
1 5 10
<210> 55
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 55
Ser Pro His Ala Phe Lys Thr Glu Ser Gly Glu Glu Thr Asp Leu
1 5 10 15
<210> 56
<211> 13
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 56
Asn Asn Leu Cys Pro Ser Gly Ser Asn Ile Ile Ser Asn
1 5 10
<210> 57
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 57
Glu Arg Pro Tyr Trp Asp Met Ser Asn Gln Asp Val Ile Asn
1 5 10
<210> 58
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 58
Val Gly Gly Thr Met Val Arg Ser Gly Gln Asp Pro Tyr Ala
1 5 10
<210> 59
<211> 13
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 59
Lys Glu Ala Leu Glu Pro Ser Gly Glu Asn Val Ile Gln
1 5 10
<210> 60
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 60
Arg Gln Arg Arg Leu Leu Gly Ser Val Gln Gln Asp Leu Glu Arg
1 5 10 15
<210> 61
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 61
Ile Asp Arg Ala Leu Asn Glu Ala Cys Glu Ser Val Ile Gln
1 5 10
<210> 62
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 62
Asn Glu Asn Asn Leu Glu Ser Ala Lys Gly Leu Leu Asp Asp Leu Arg
1 5 10 15
<210> 63
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 63
Asp Ser Lys Ser Leu Arg Thr Ala Leu Gln Lys Glu Ile Thr Thr Arg
1 5 10 15
<210> 64
<211> 13
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 64
Thr Gly Gly Asp Ile Asn Ala Ala Ile Glu Arg Leu Leu
1 5 10
<210> 65
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 65
Ser Leu Asp Asn Leu Lys Ala Ser Val Ser Gln Val Glu Ala Asp Leu
1 5 10 15
<210> 66
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 66
Asp Glu Arg Arg Phe Lys Ala Ala Asp Leu Asn Gly Asp Leu
1 5 10
<210> 67
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 67
Lys Pro Ala Pro Ala Leu Arg Ser Ala Arg Ser Ala Pro Glu Asn
1 5 10 15
<210> 68
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 68
Arg Thr Ser Tyr Val Thr Ser Val Glu Glu Asn Thr Val Asp
1 5 10
<210> 69
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 69
Arg Ala Val Glu Phe Gln Glu Ala Gln Ala Tyr Ala Asp Asp Asn
1 5 10 15
<210> 70
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 70
Arg Asp Leu Ala Gln Tyr Asp Ala Ala His His Glu Glu Phe
1 5 10
<210> 71
<211> 13
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 71
Gly Gln Gln Trp Thr Tyr Glu Gln Arg Lys Ile Val Glu
1 5 10
<210> 72
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 72
Glu Gly Glu Tyr Gln Gly Ile Pro Arg Ala Glu Ser Gly Gly
1 5 10
<210> 73
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 73
Ser Pro Glu Glu Phe Asp Glu Val Ser Arg Ile Val Gly Ser Val Glu
1 5 10 15
<210> 74
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 74
Ile Ala Gly Glu Trp Gln Val Leu His Arg Glu Gly Ala Ile Thr
1 5 10 15
<210> 75
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 75
Glu Pro Ala Glu Phe Ile Ile Asp Thr Arg Asp Ala Gly Tyr Gly
1 5 10 15
<210> 76
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 76
Asp Leu Glu Glu Leu Glu Val Leu Glu Arg Lys Pro Ala Ala Gly
1 5 10 15
<210> 77
<211> 16
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 77
Gly Met Asn Ile Val Glu Ala Met Glu Arg Phe Gly Ser Arg Asn Gly
1 5 10 15
<210> 78
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 78
Gly Phe Asn Trp Asn Trp Ile Asn Lys Gln Gln Gly Lys Arg Gly
1 5 10 15
<210> 79
<211> 13
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 79
Gly Pro Phe Ser Phe Ser Val Ile Asp Lys Pro Pro Gly
1 5 10
<210> 80
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 80
Leu Asn Glu Leu Lys Pro Ile Ser Lys Gly Gly His Ser Ser
1 5 10
<210> 81
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 81
Asp Val Asn Glu Tyr Ala Pro Val Phe Lys Glu Lys Ser Tyr Lys
1 5 10 15
<210> 82
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 82
Leu Ala Pro Thr Trp Glu Glu Leu Ser Lys Lys Glu Phe Pro Gly
1 5 10 15
<210> 83
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 83
Thr Arg Asn Glu Val Ile Pro Met Ser His Pro Gly Ala Val Asp
1 5 10 15
<210> 84
<211> 14
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 84
Glu Asn Pro Tyr Phe Ala Pro Asn Pro Lys Ile Ile Arg Gln
1 5 10
<210> 85
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 85
Tyr Glu Asp Lys Phe Arg Asn Asn Leu Lys Gly Lys Arg Leu Asp
1 5 10 15
<210> 86
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 86
Lys Asn Gly Ala Tyr Lys Val Glu Thr Lys Lys Tyr Asp Phe Tyr
1 5 10 15
<210> 87
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 87
Gly Glu Gly Leu Phe Gln Pro Ala His Arg Tyr Pro Asp Ala Gly
1 5 10 15
<210> 88
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 88
Arg Asn Gln Trp Lys Cys Leu Gly Lys Pro Val Gly Ala Glu Met
1 5 10 15
<210> 89
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 89
Val Pro Ala Trp Thr Arg Ala Trp Arg Asn Ser Ser Pro Lys Gly
1 5 10 15
<210> 90
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 90
Leu His Pro Glu Leu Leu Pro Leu Trp Arg Leu Leu Pro Asp Gly
1 5 10 15
<210> 91
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 91
Asp Gly Gly Ser Tyr Phe Ser Leu Trp Lys Ile Trp Thr Gln Val
1 5 10 15
<210> 92
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 92
His Glu Gly Gly Phe Pro Pro Leu Leu Arg Arg Ala Ala Glu Asp
1 5 10 15
<210> 93
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 93
Leu Ser Pro Val Trp Cys Leu Gln Trp Lys Leu Ser Gly Thr Asp
1 5 10 15
<210> 94
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 94
Lys Asn Thr Ile Val Tyr Thr Thr Lys Gln Val Gln Ser Cys Gln
1 5 10 15
<210> 95
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 95
Asn Asn Glu Gln Phe Gln Trp Lys Ile Arg His Val Gly Pro Glu
1 5 10 15
<210> 96
<211> 15
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Описание искусственной последовательности: синтетический пептид
<400> 96
Tyr Lys Gly Gly Tyr Glu Leu Val Lys Lys Ser Gln Thr Glu Leu
1 5 10 15
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ИДЕНТИФИКАЦИЯ, ПРОИЗВОДСТВО И ПРИМЕНЕНИЕ НЕОАНТИГЕНОВ | 2016 |
|
RU2729116C2 |
| НЕОАНТИГЕННЫЕ ВЕКТОРЫ НА ОСНОВЕ АЛЬФАВИРУСА | 2018 |
|
RU2803566C2 |
| ГЕНЕТИЧЕСКИ МОДИФИЦИРОВАННЫЕ МЫШИ, ЭКСПРЕССИРУЮЩИЕ ХИМЕРНЫЕ МОЛЕКУЛЫ ГЛАВНОГО КОМПЛЕКСА ГИСТОСОВМЕСТИМОСТИ | 2012 |
|
RU2797549C2 |
| МОЛОЧНАЯ СМЕСЬ СО СПЕЦИФИЧЕСКИМИ ПЕПТИДАМИ БЕТА-ЛАКТОГЛОБУЛИНА | 2019 |
|
RU2780747C2 |
| НЕОАНТИГЕННЫЕ КОМПОЗИЦИИ И ИХ ИСПОЛЬЗОВАНИЕ | 2020 |
|
RU2832574C2 |
| ГЕНЕТИЧЕСКИ МОДИФИЦИРОВАННЫЕ МЫШИ, ЭКСПРЕССИРУЮЩИЕ ХИМЕРНЫЕ МОЛЕКУЛЫ ГЛАВНОГО КОМПЛЕКСА ГИСТОСОВМЕСТИМОСТИ | 2012 |
|
RU2660564C2 |
| СПОСОБЫ ПРОГНОЗИРОВАНИЯ ПРИМЕНИМОСТИ СПЕЦИФИЧНЫХ ДЛЯ ЗАБОЛЕВАНИЯ АМИНОКИСЛОТНЫХ МОДИФИКАЦИЙ ДЛЯ ИММУНОТЕРАПИИ | 2018 |
|
RU2799341C2 |
| RA АНТИГЕННЫЕ ПЕПТИДЫ | 2004 |
|
RU2359974C2 |
| СПОСОБЫ ЛЕЧЕНИЯ АУТОИММУННОГО ЗАБОЛЕВАНИЯ С ИСПОЛЬЗОВАНИЕМ АЛЛОГЕННЫХ Т-КЛЕТОК | 2017 |
|
RU2773831C2 |
| ПРОФИЛАКТИЧЕСКАЯ ИЛИ ТЕРАПЕВТИЧЕСКАЯ ПОЛИЭПИТОПНАЯ ПРОТИВОТУБЕРКУЛЕЗНАЯ ВАКЦИННАЯ КОНСТРУКЦИЯ, ОБЕСПЕЧИВАЮЩАЯ ИНДУКЦИЮ КЛЕТОЧНОГО ИММУННОГО ОТВЕТА CD4+ ИЛИ CD8+ Т-ЛИМФОЦИТОВ | 2011 |
|
RU2539035C2 |
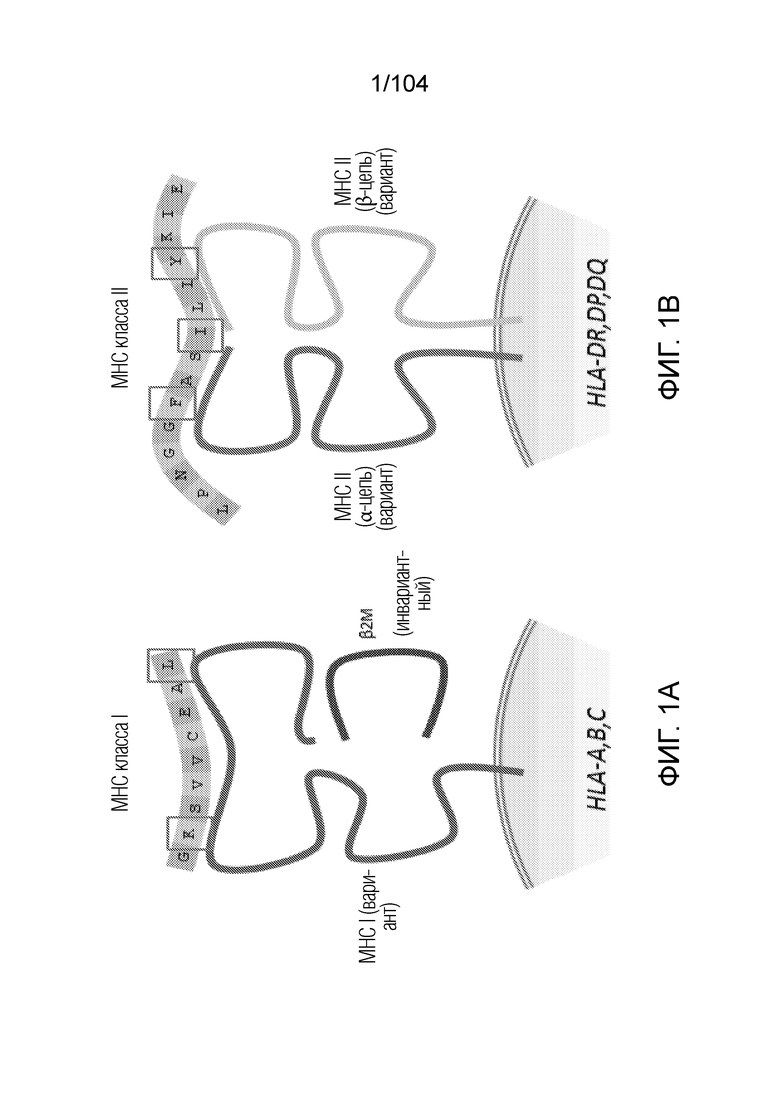
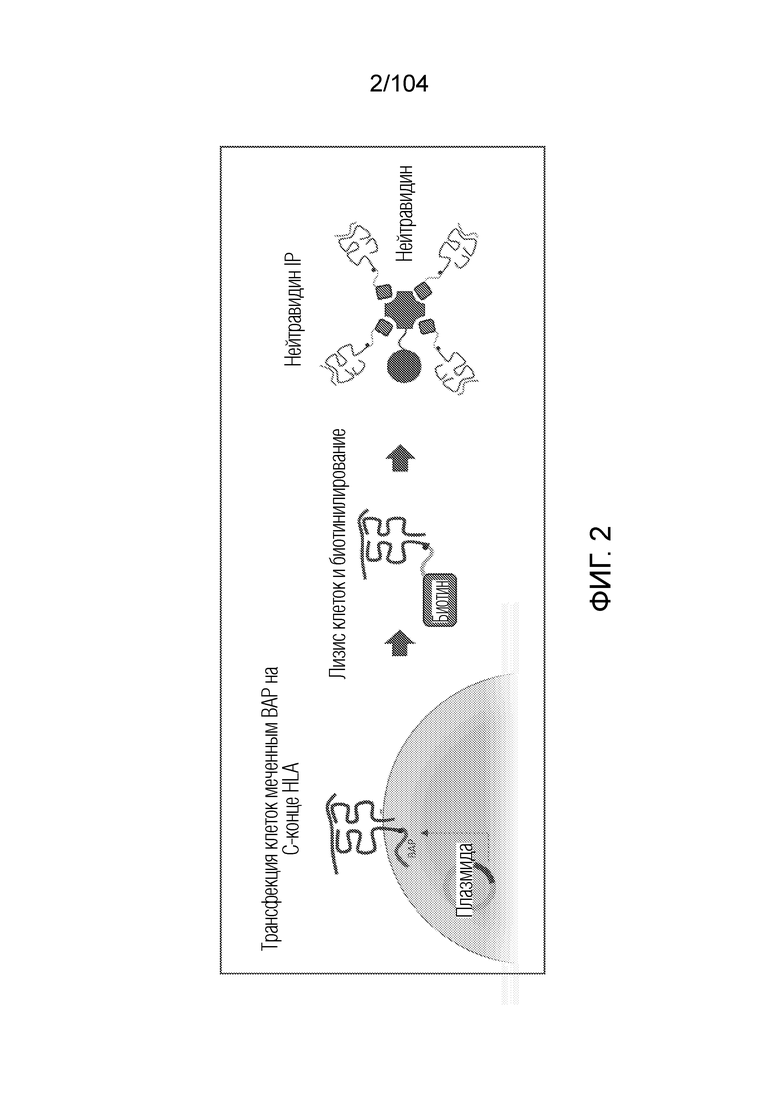
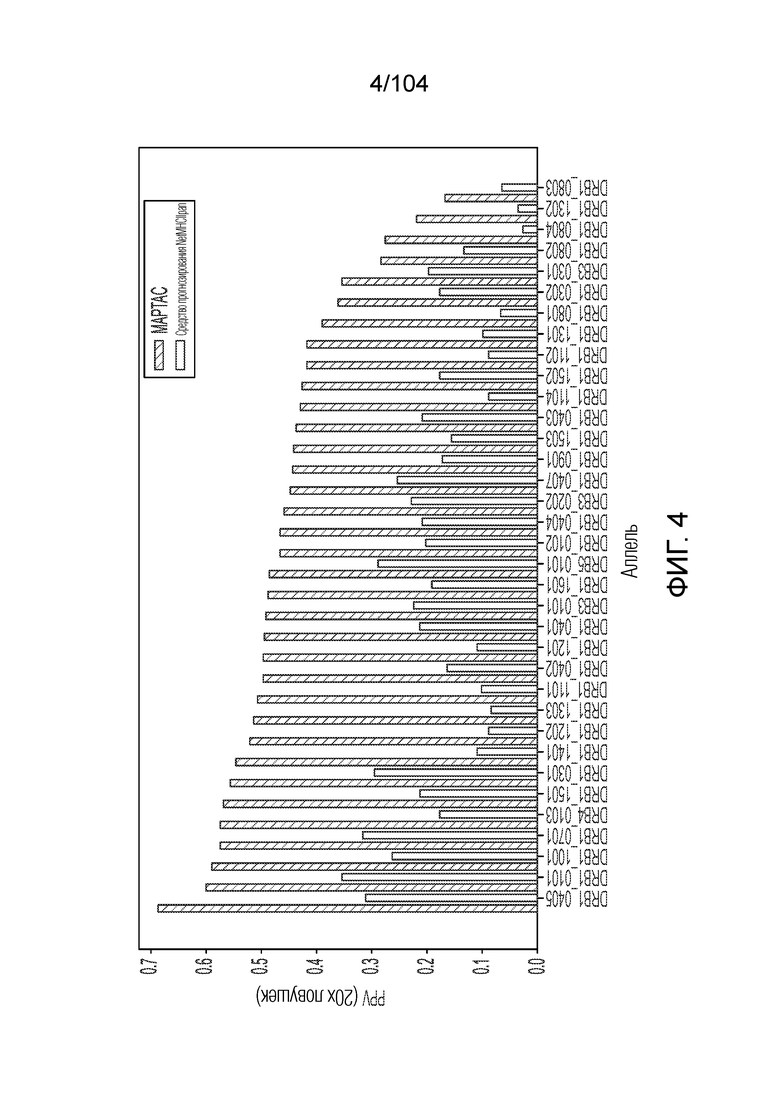
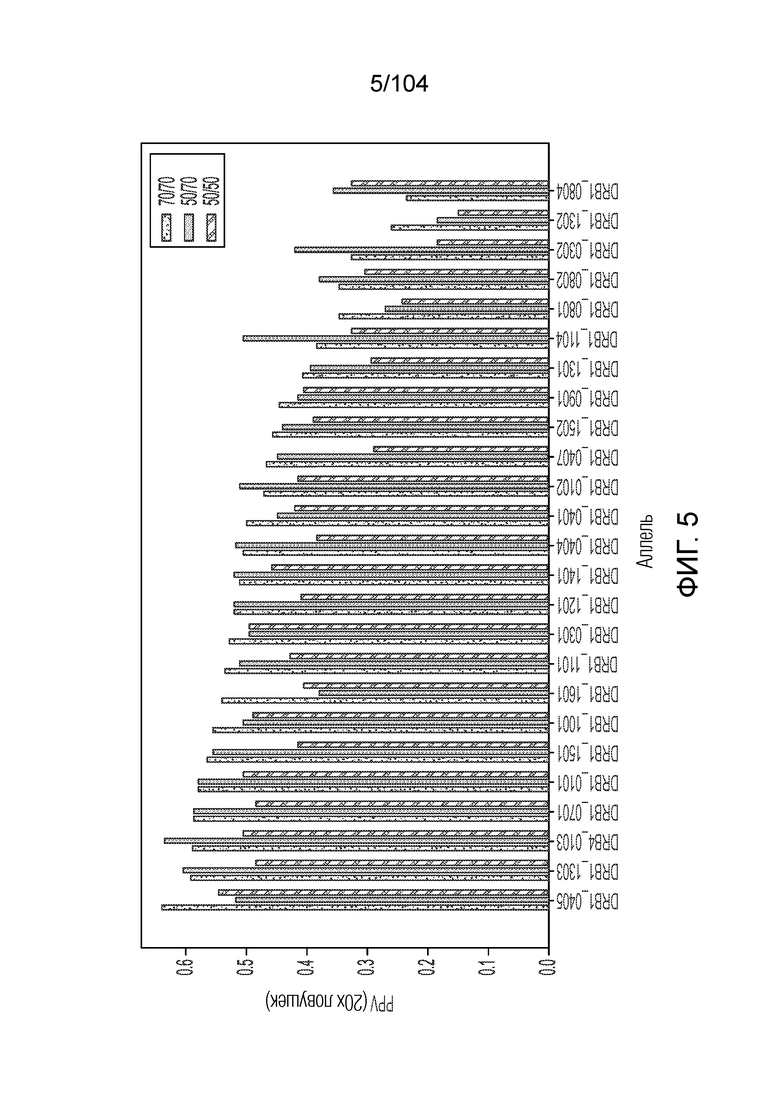
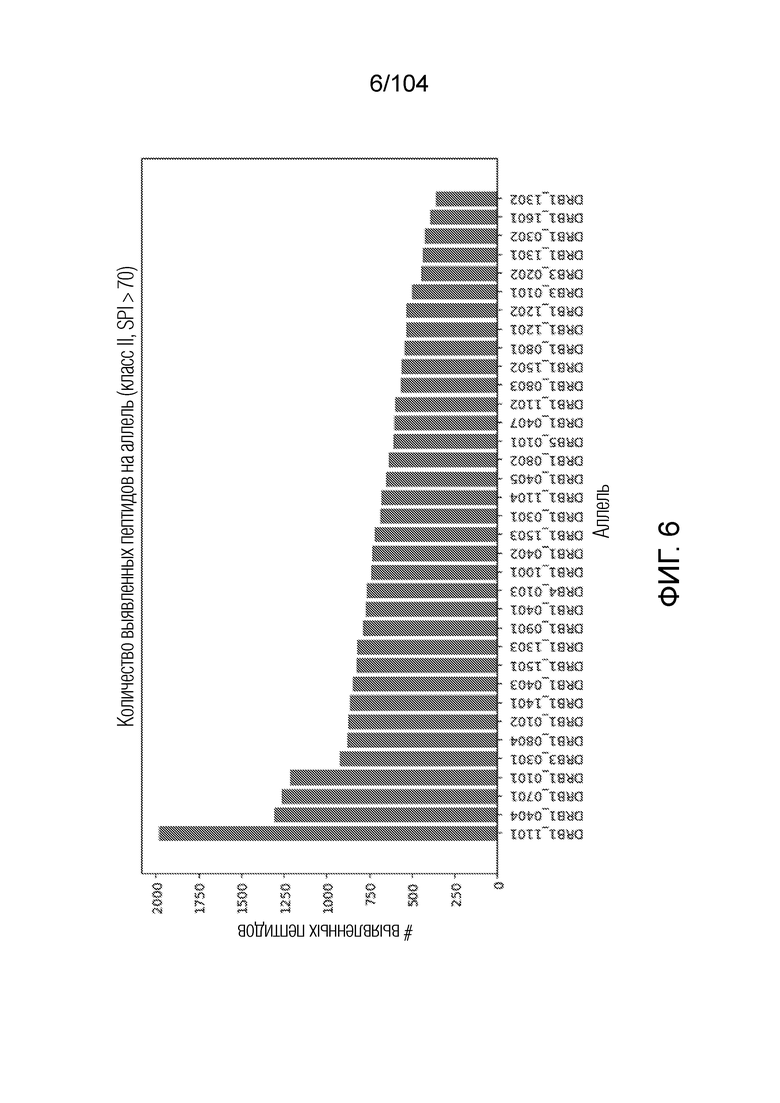
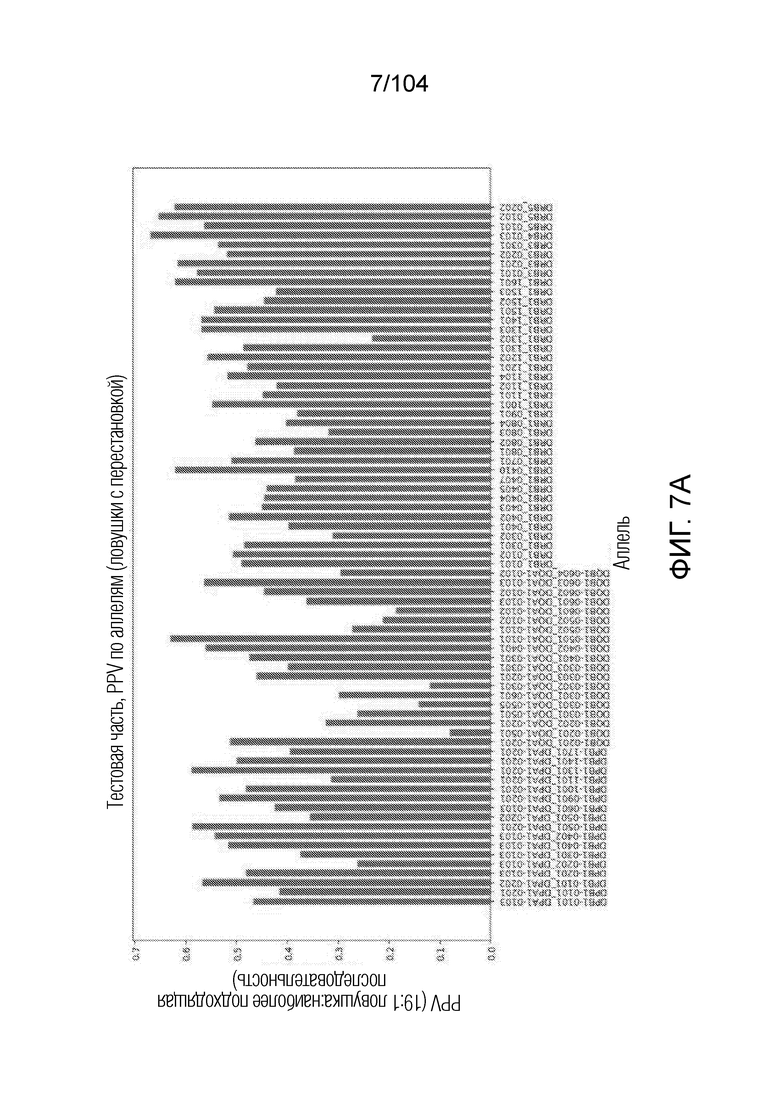
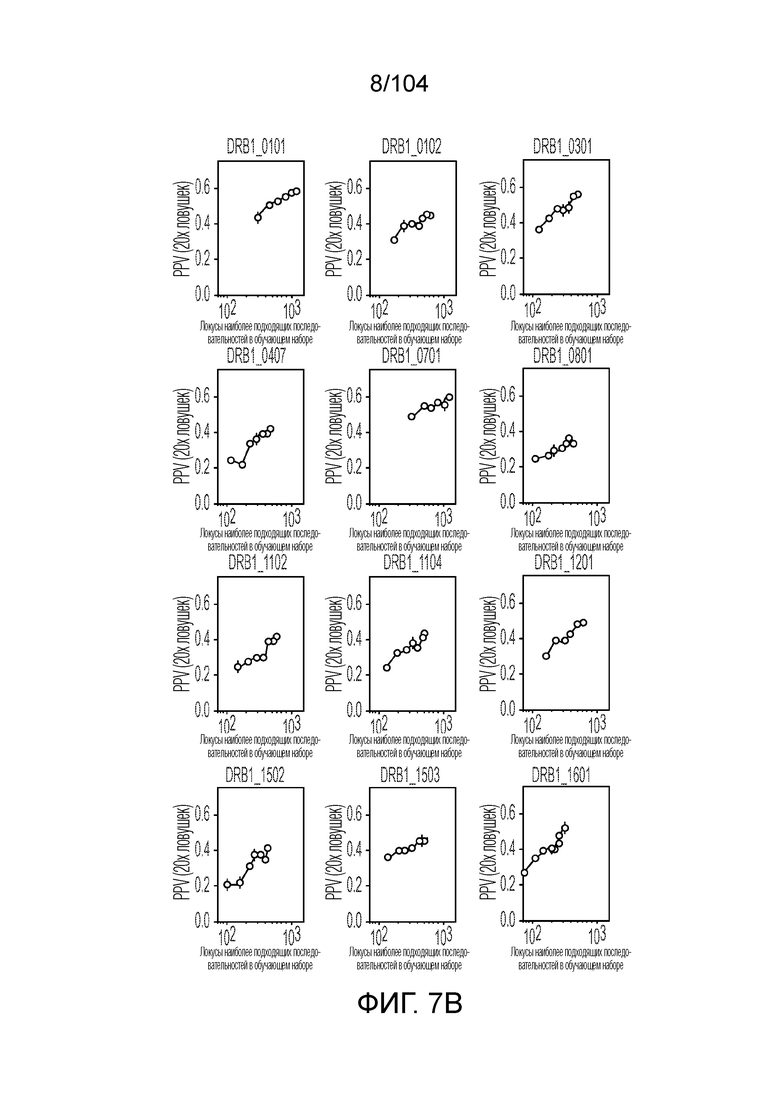
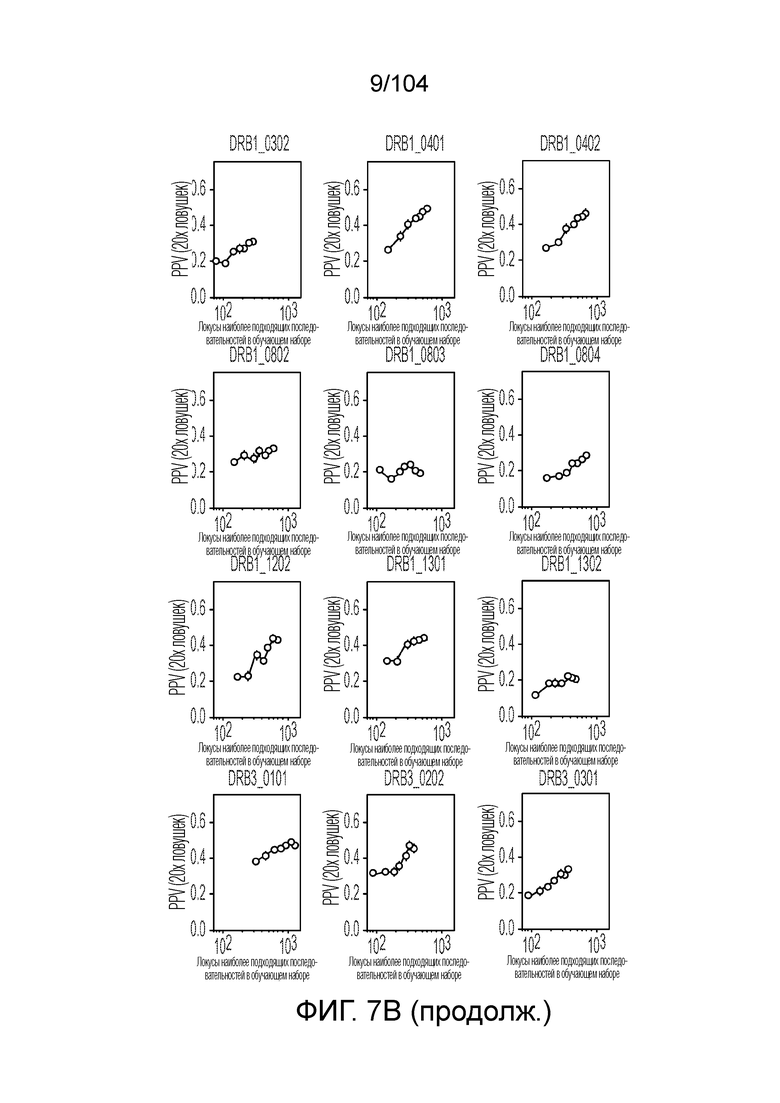

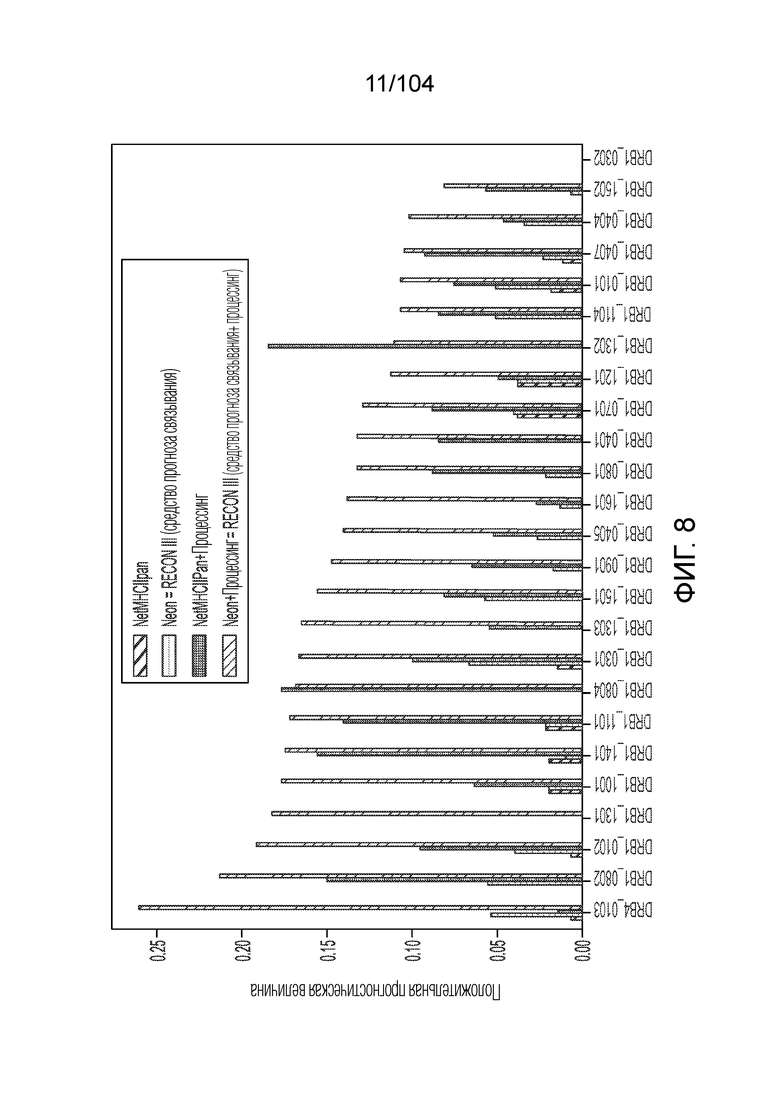
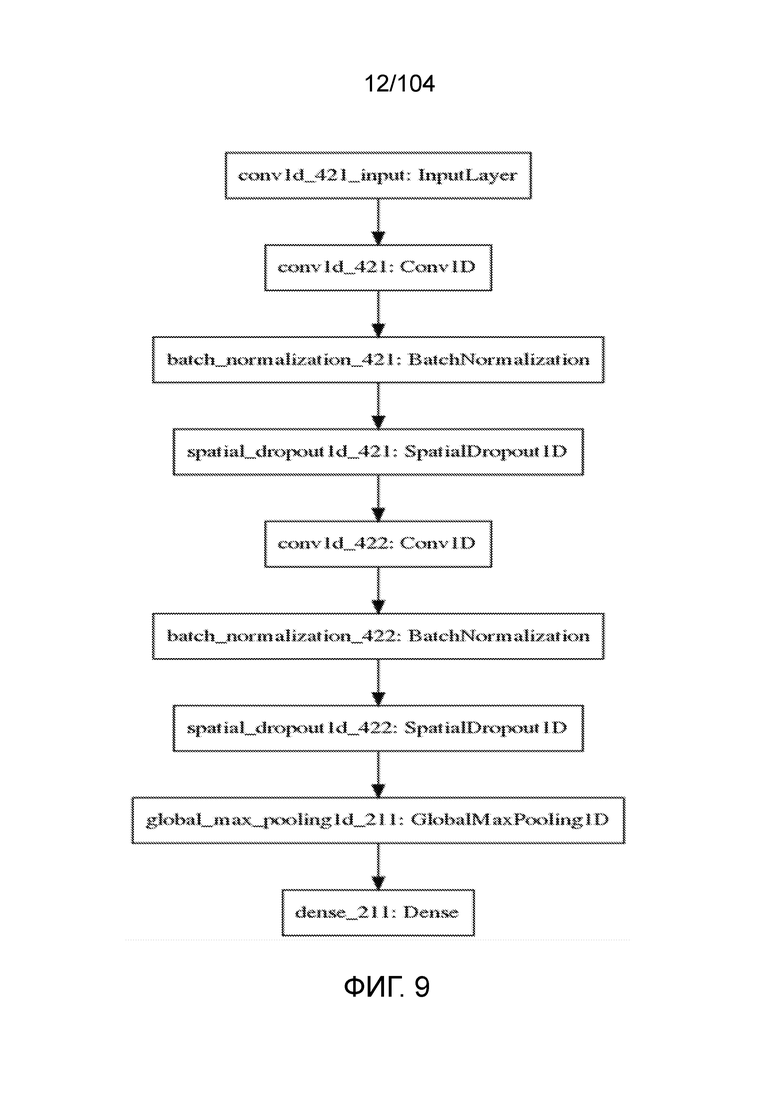
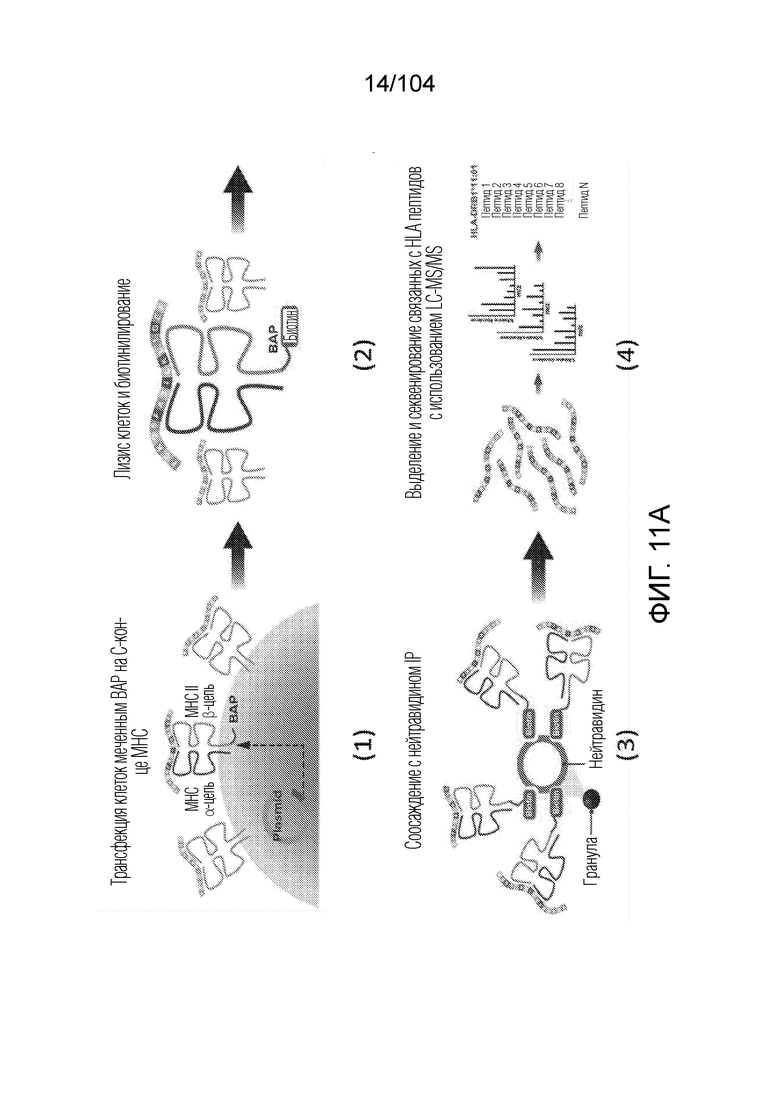
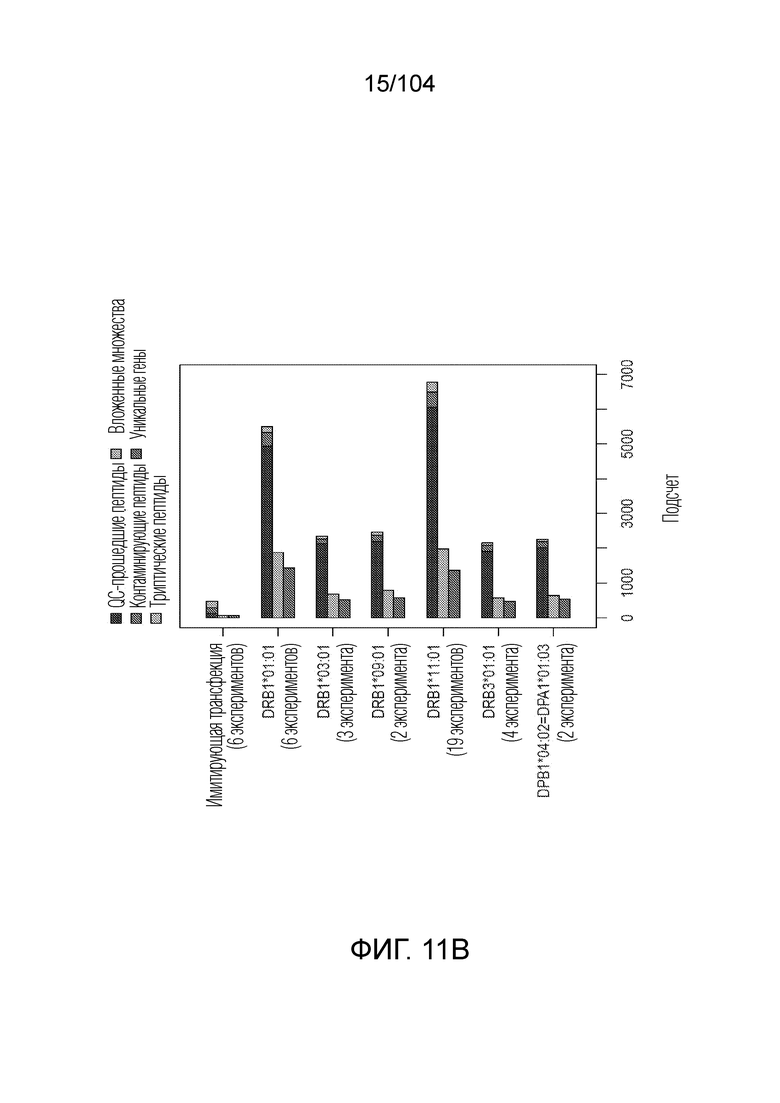
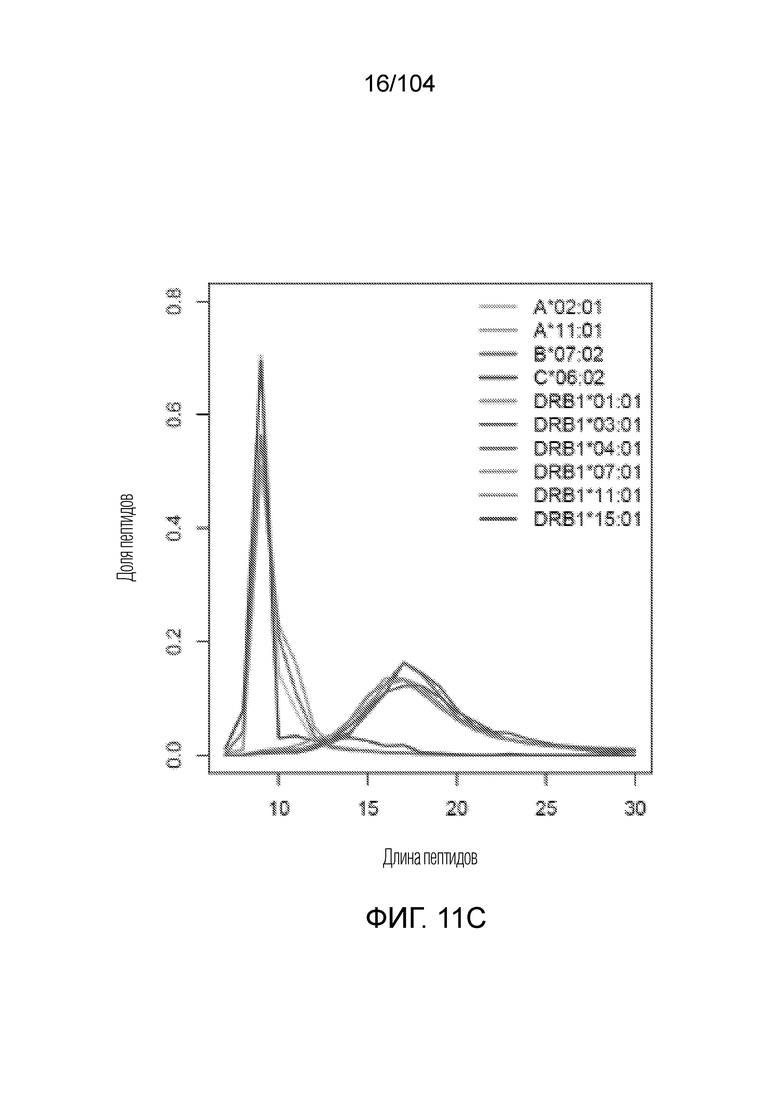



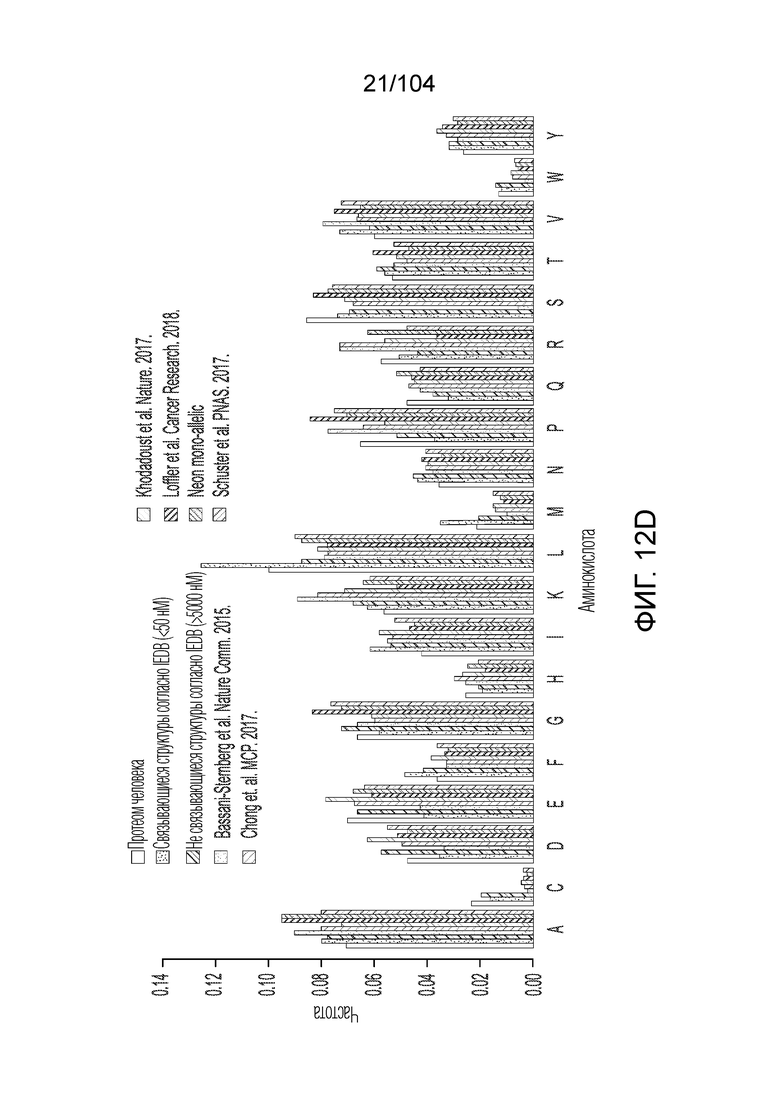

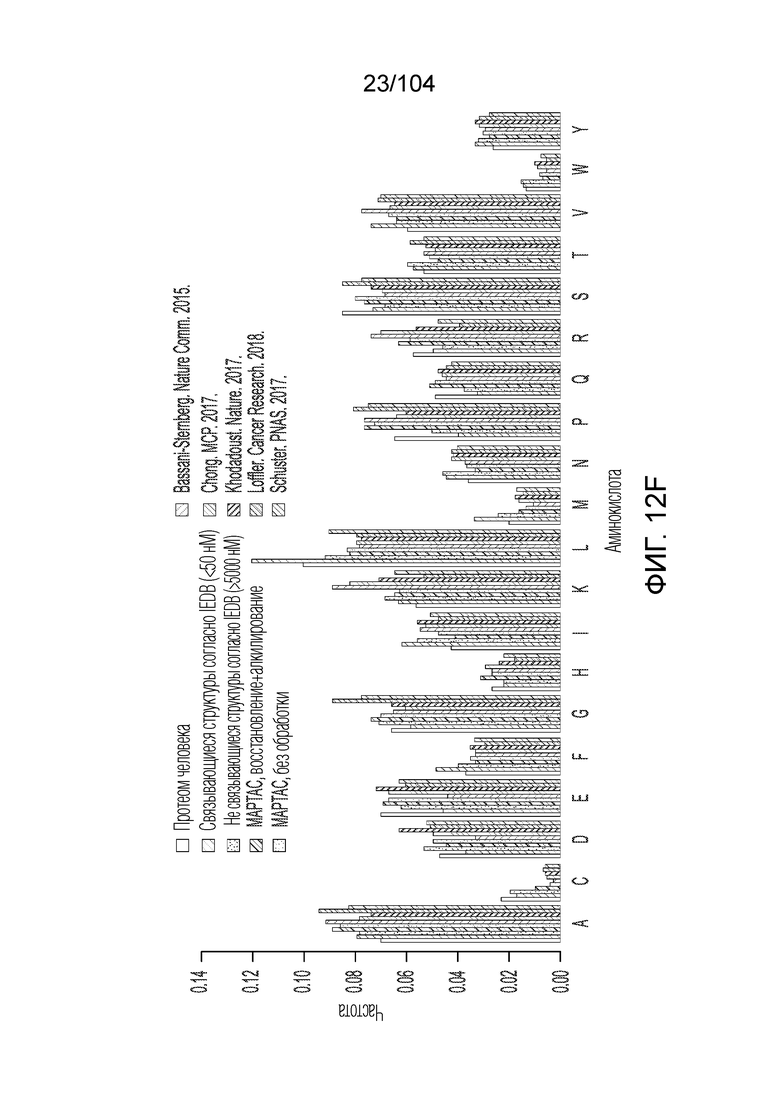


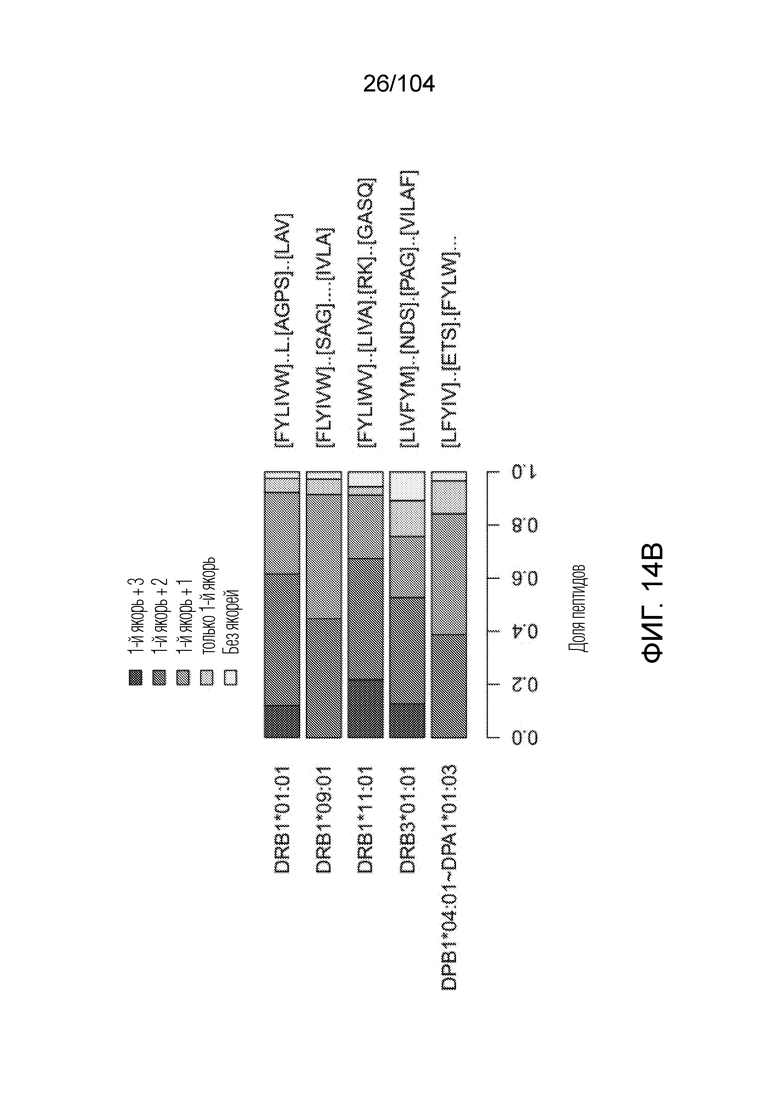

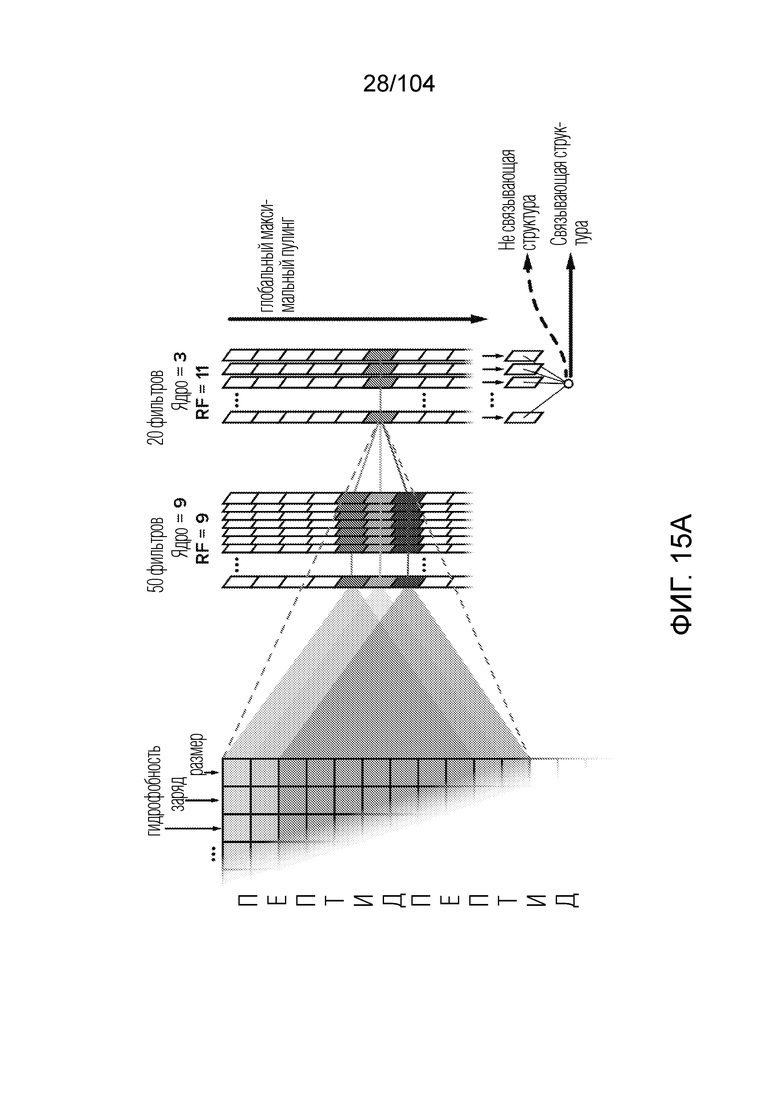
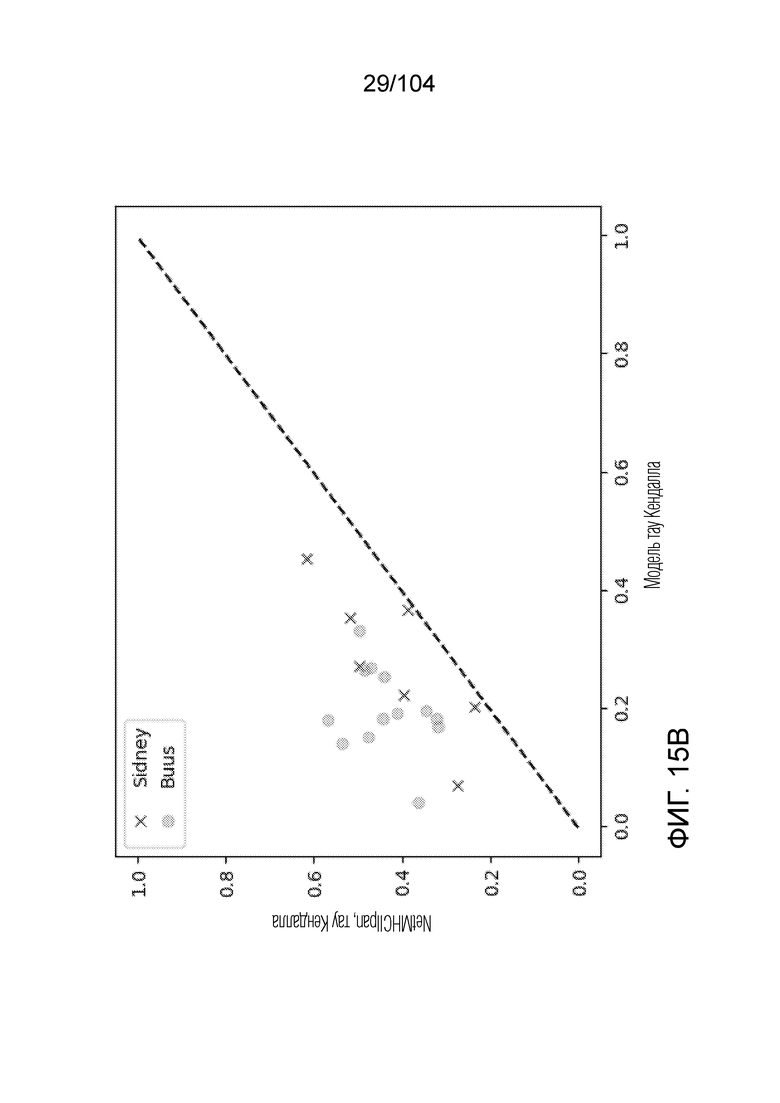



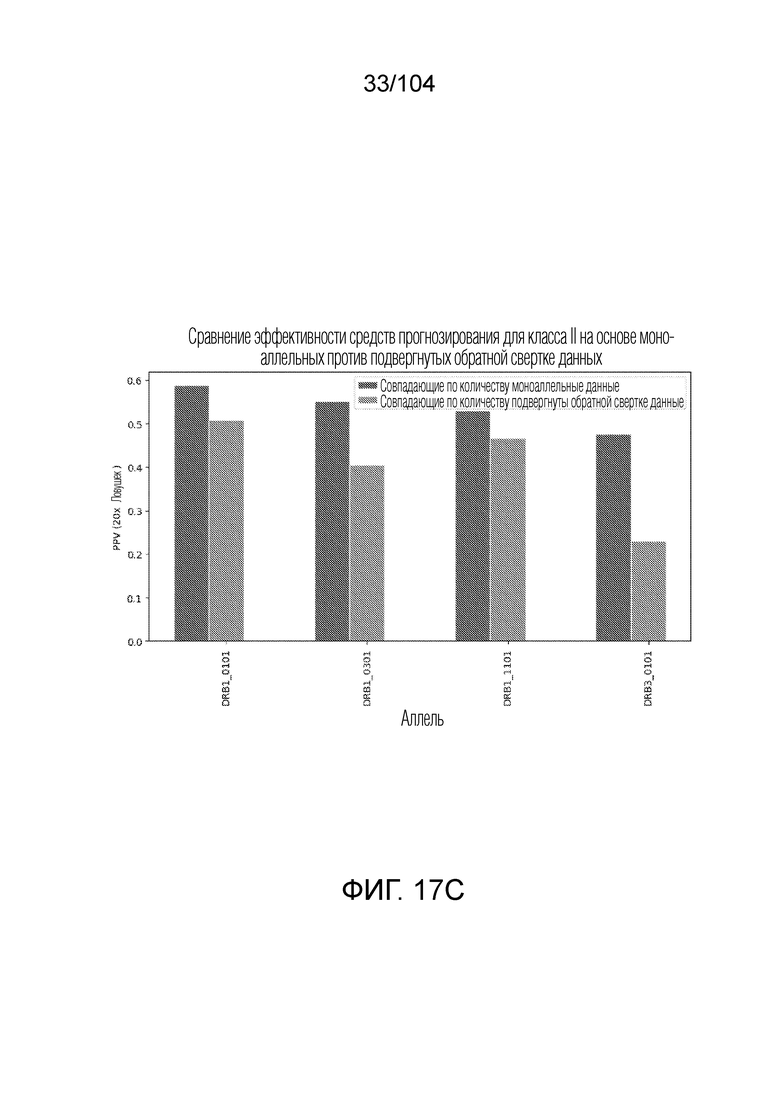

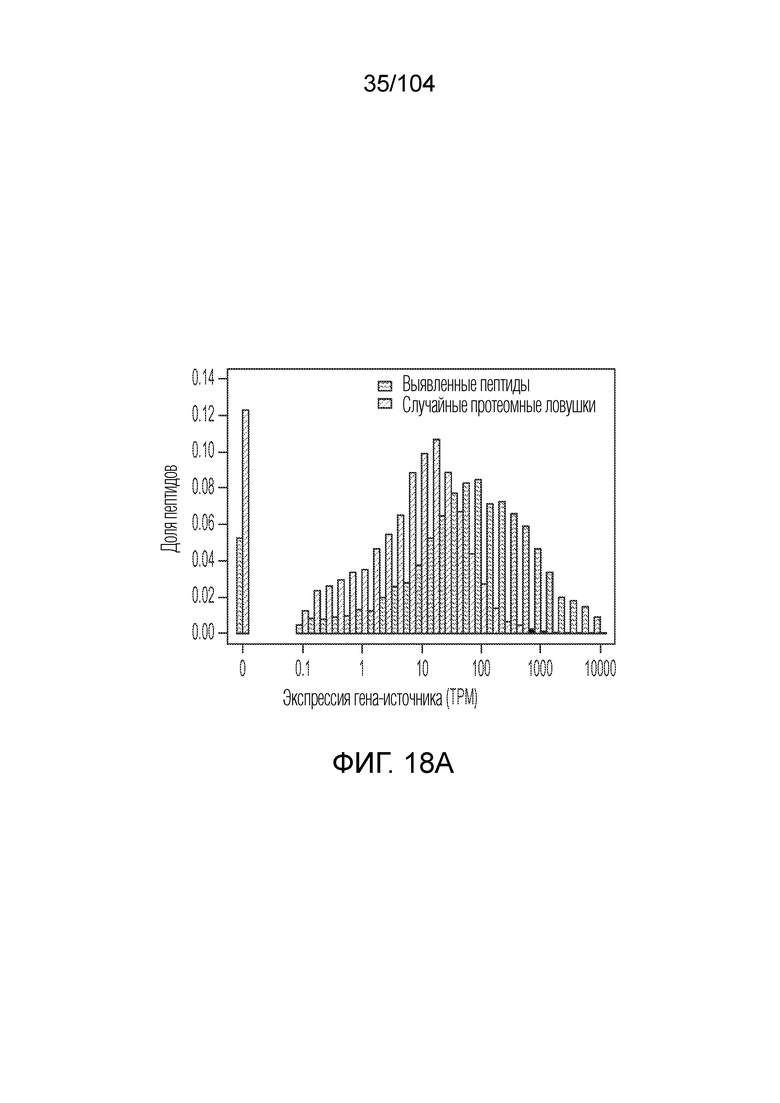
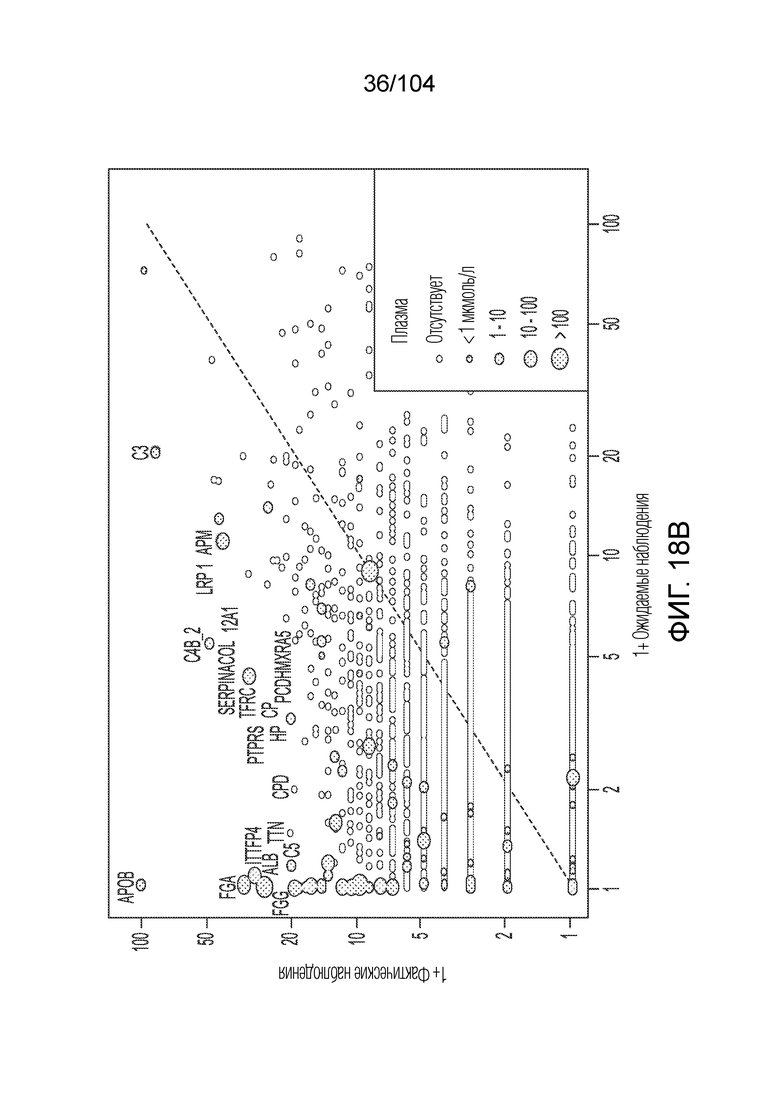
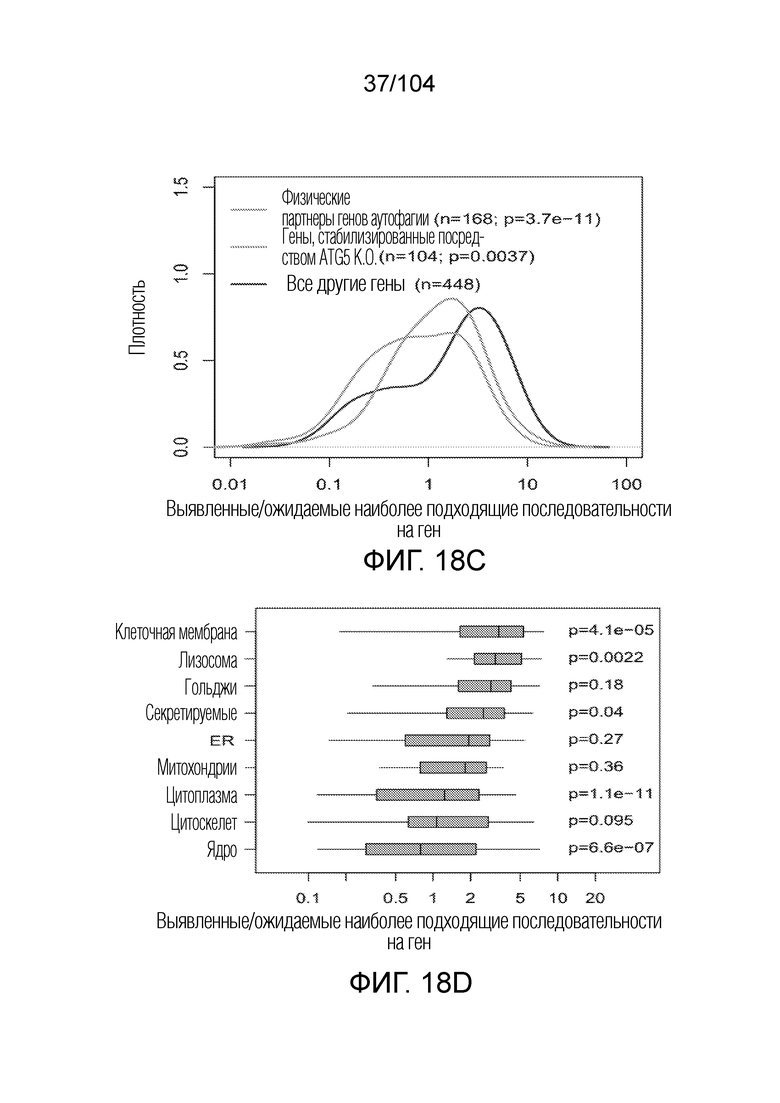
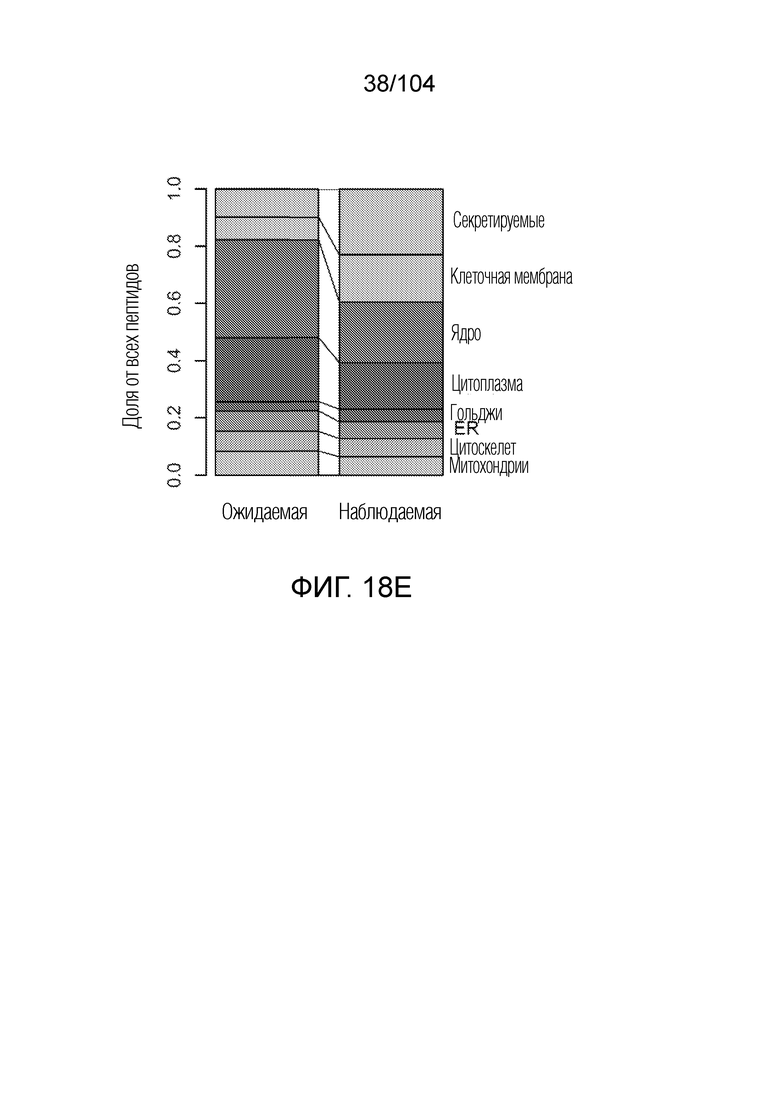
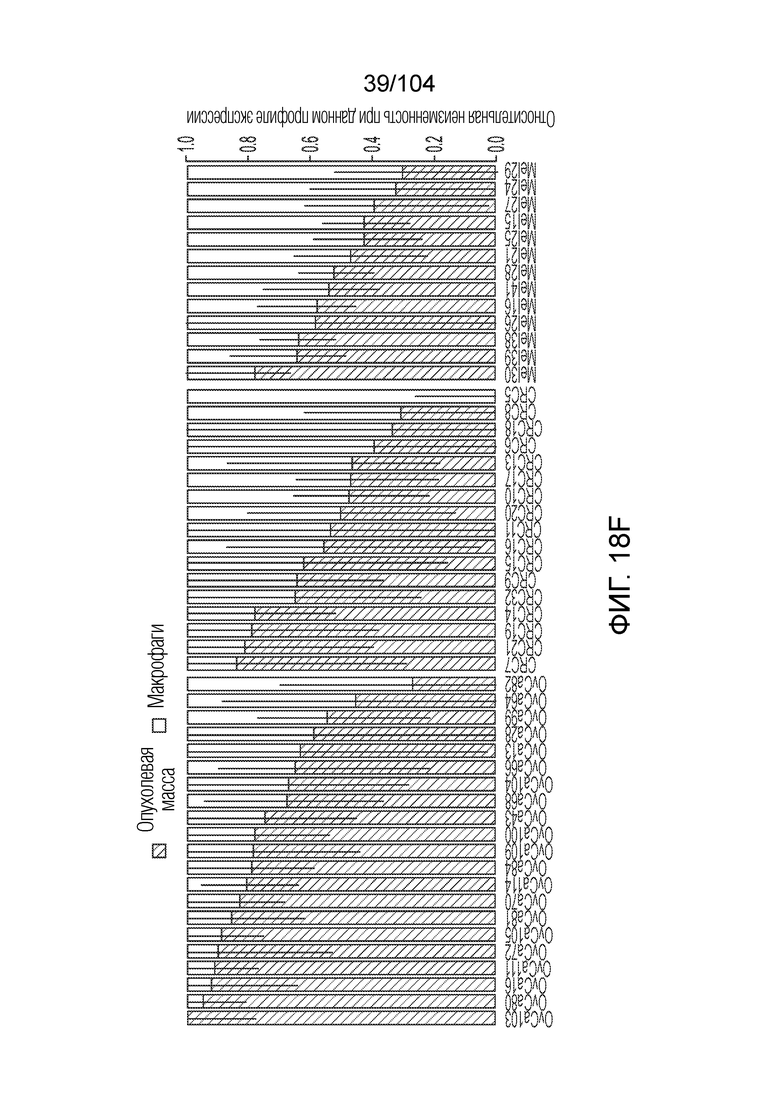
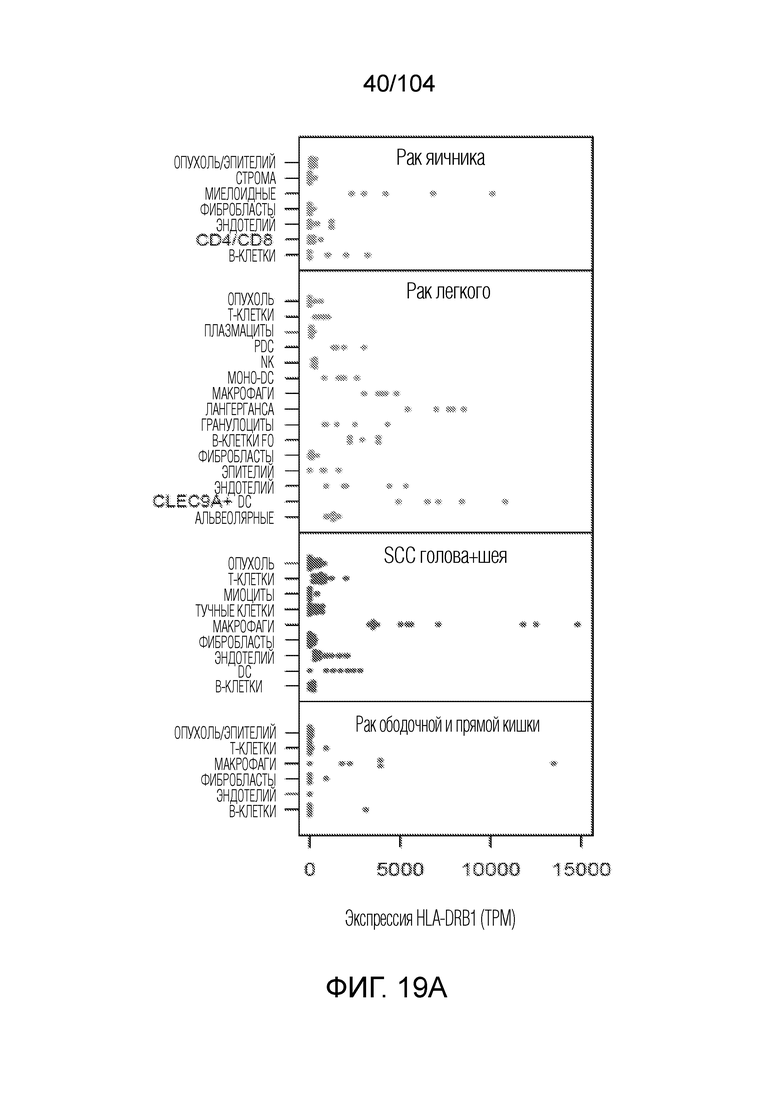
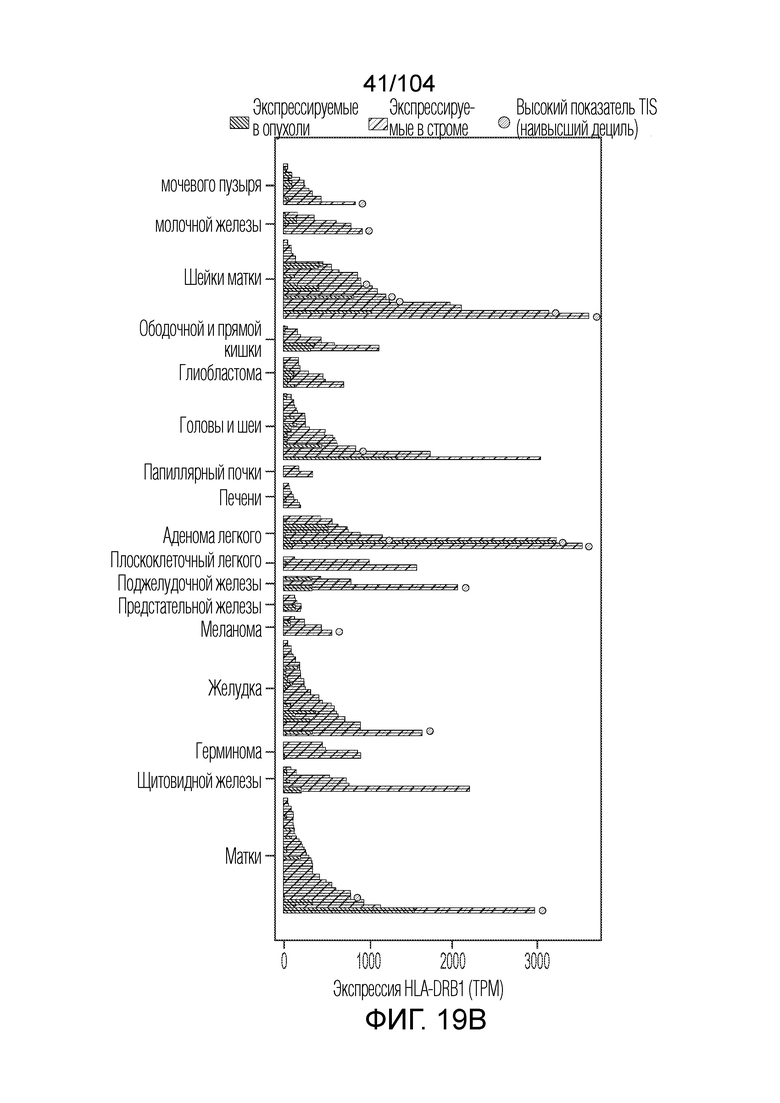
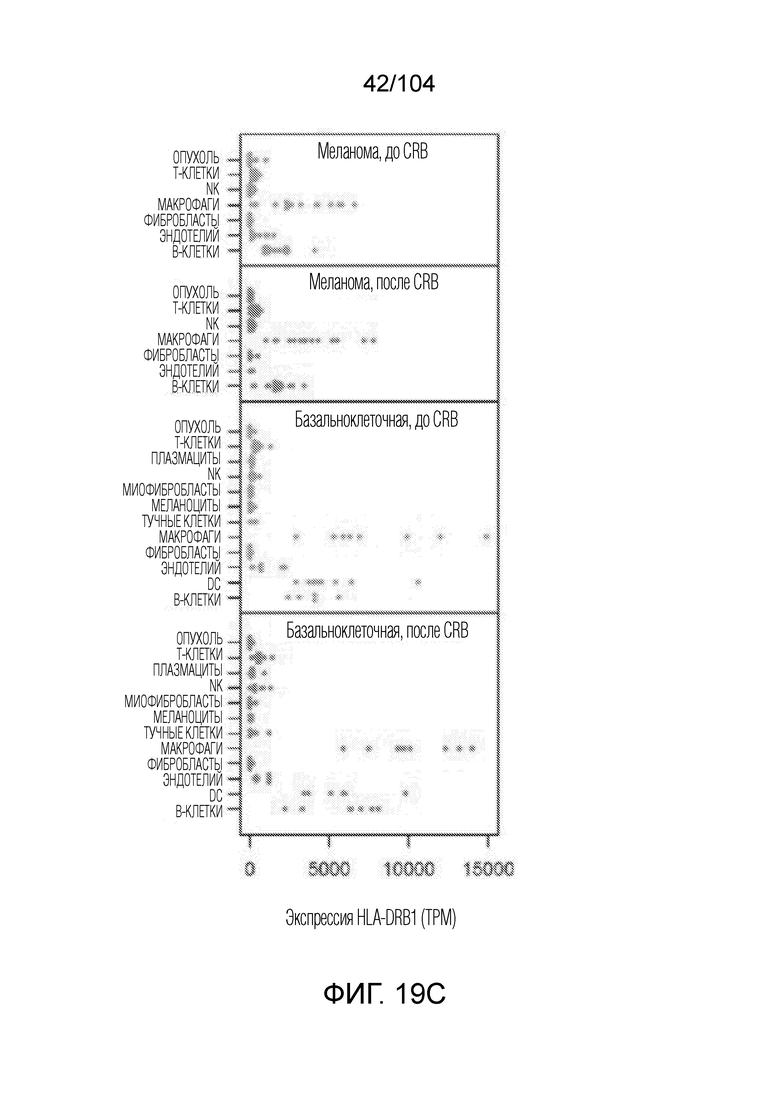
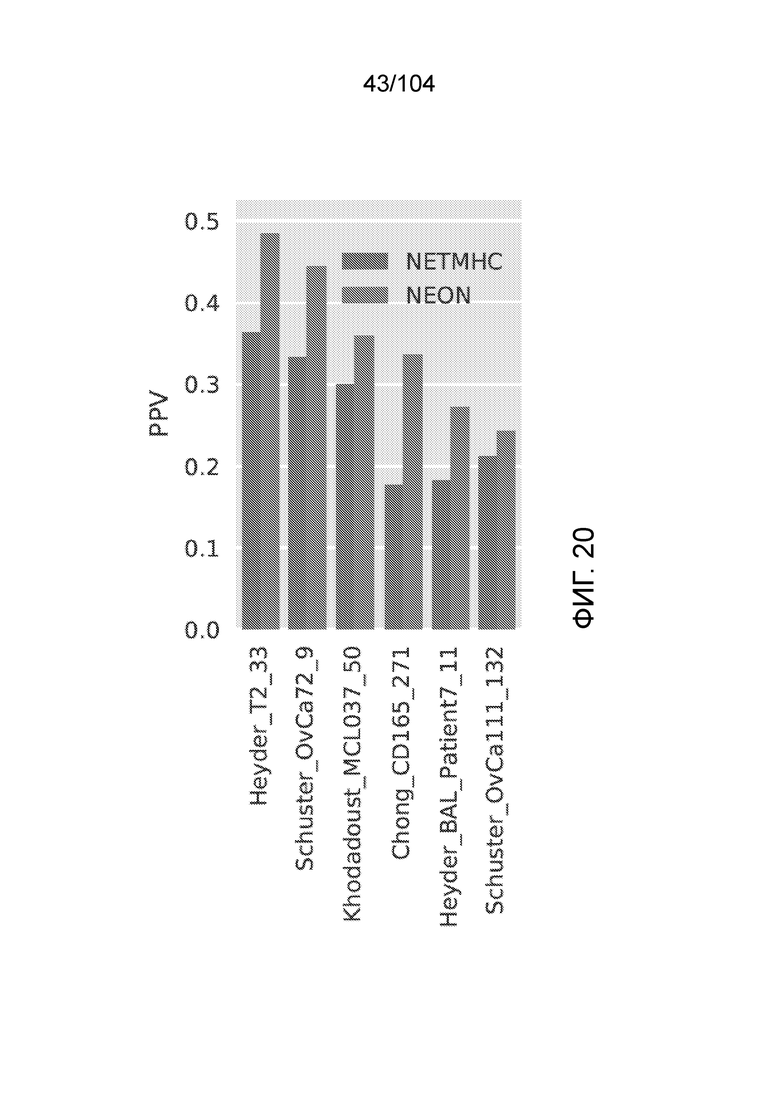
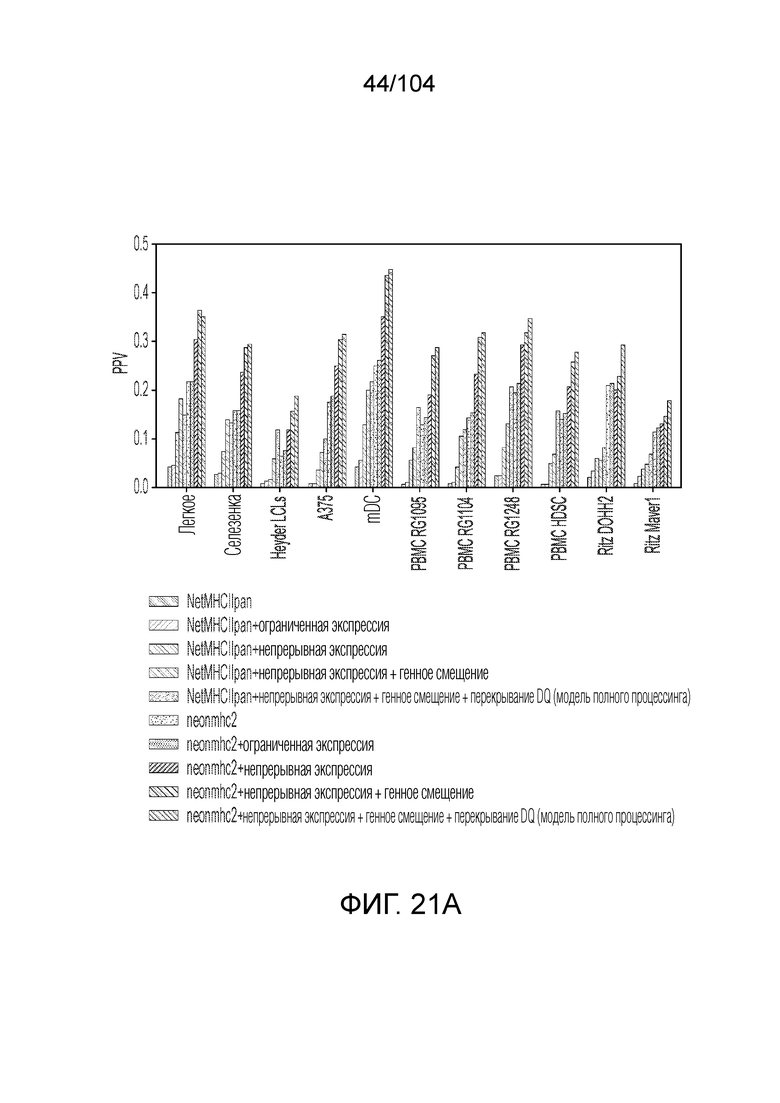
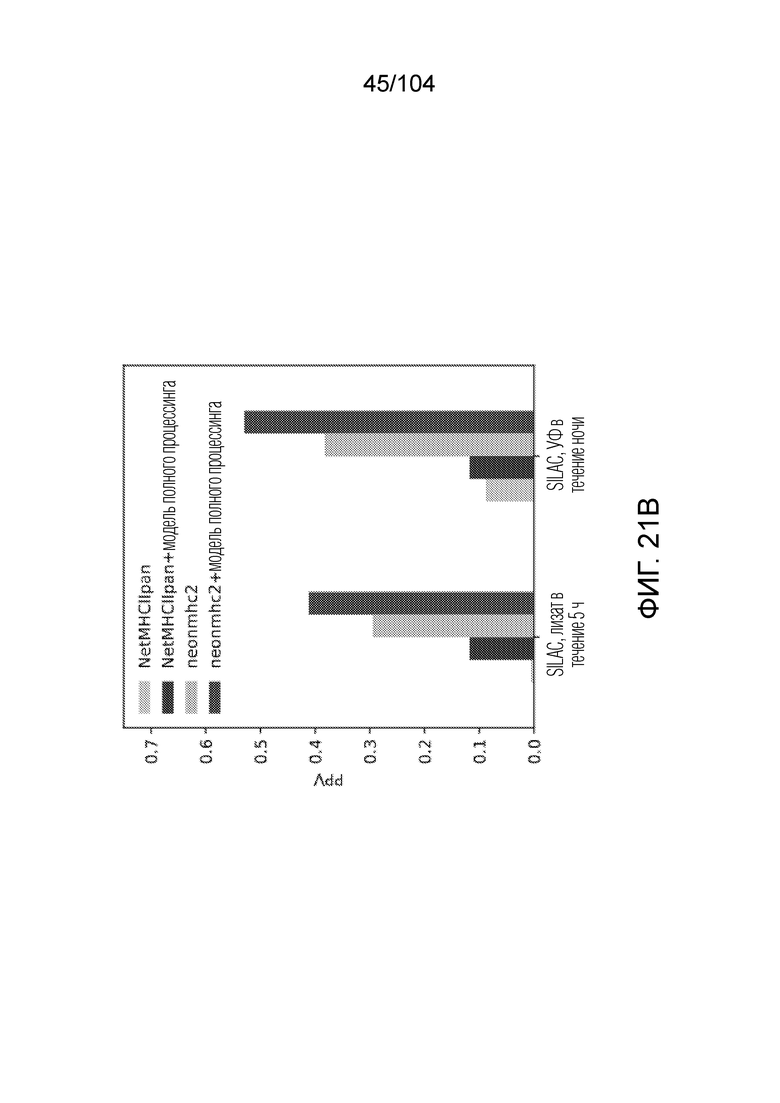
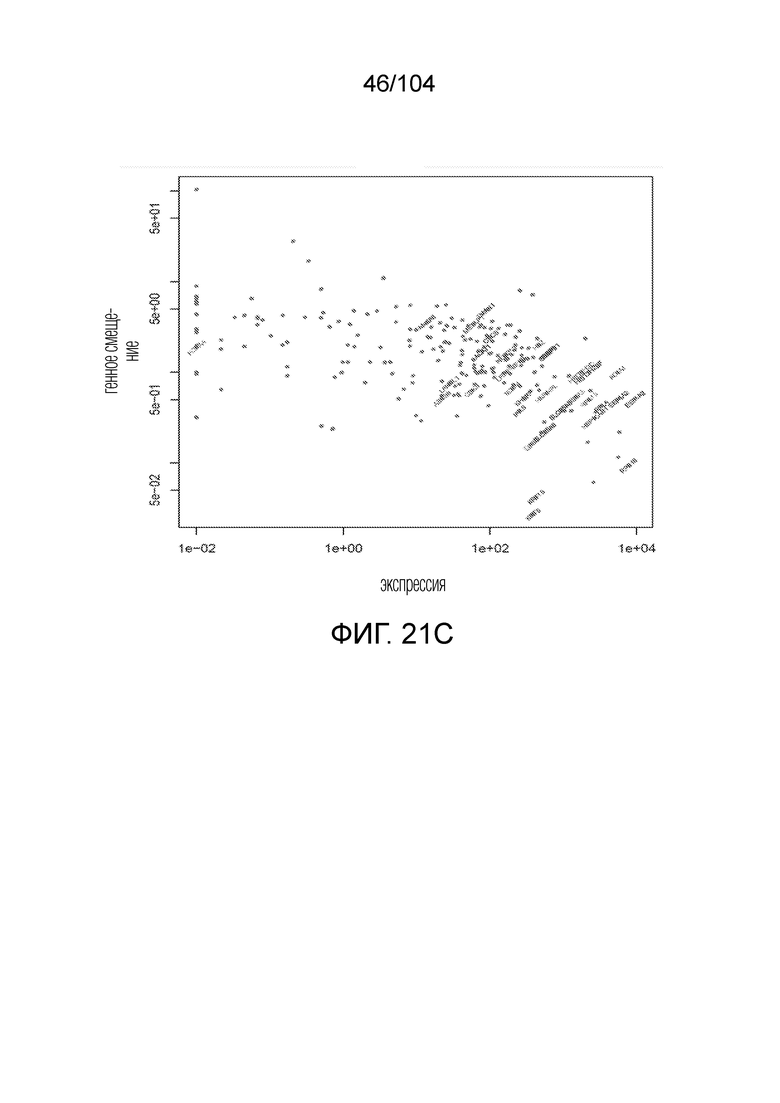
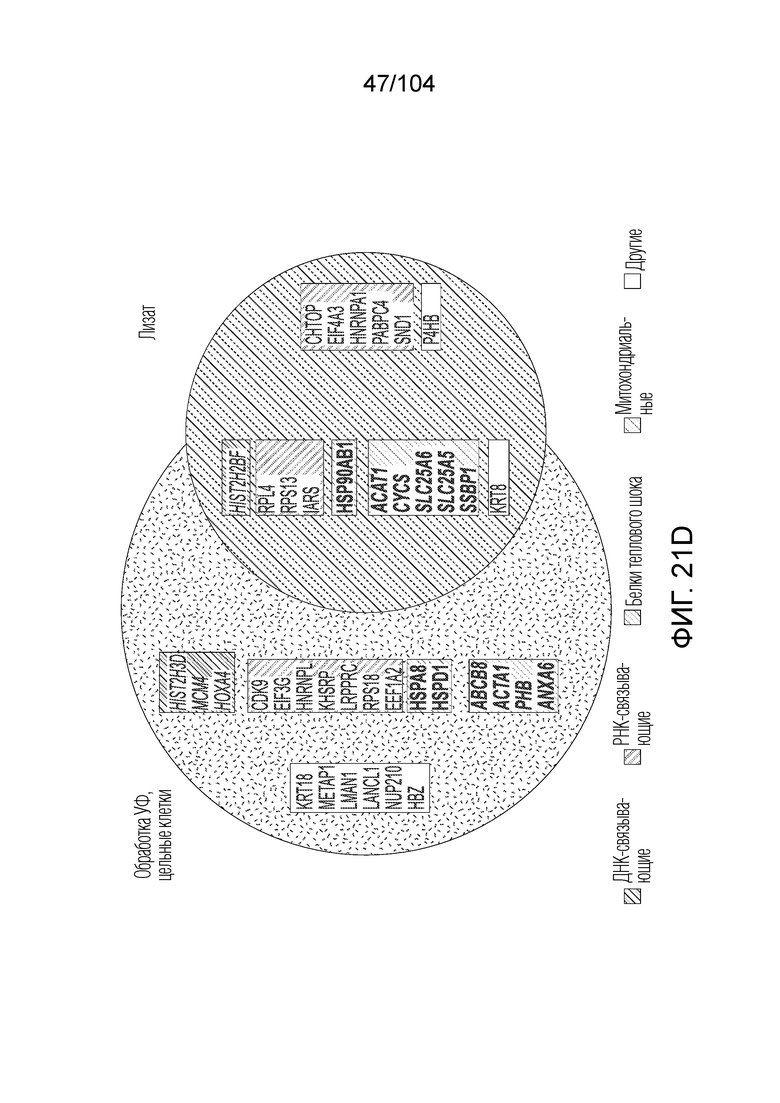
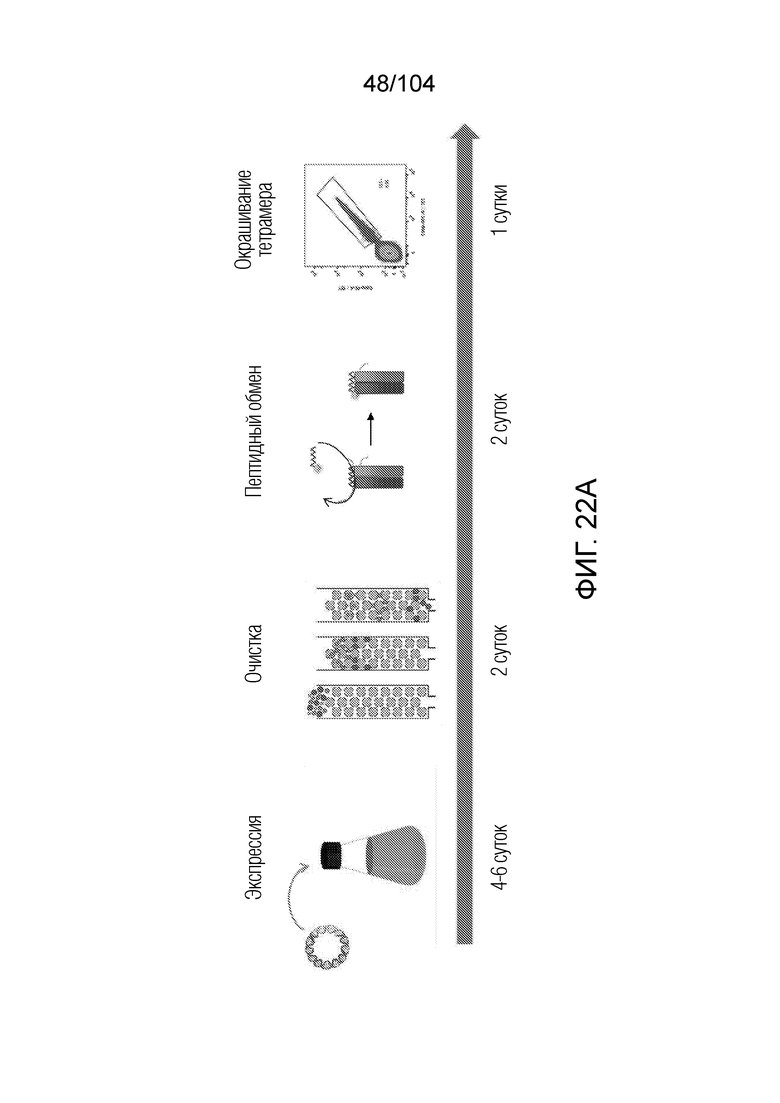
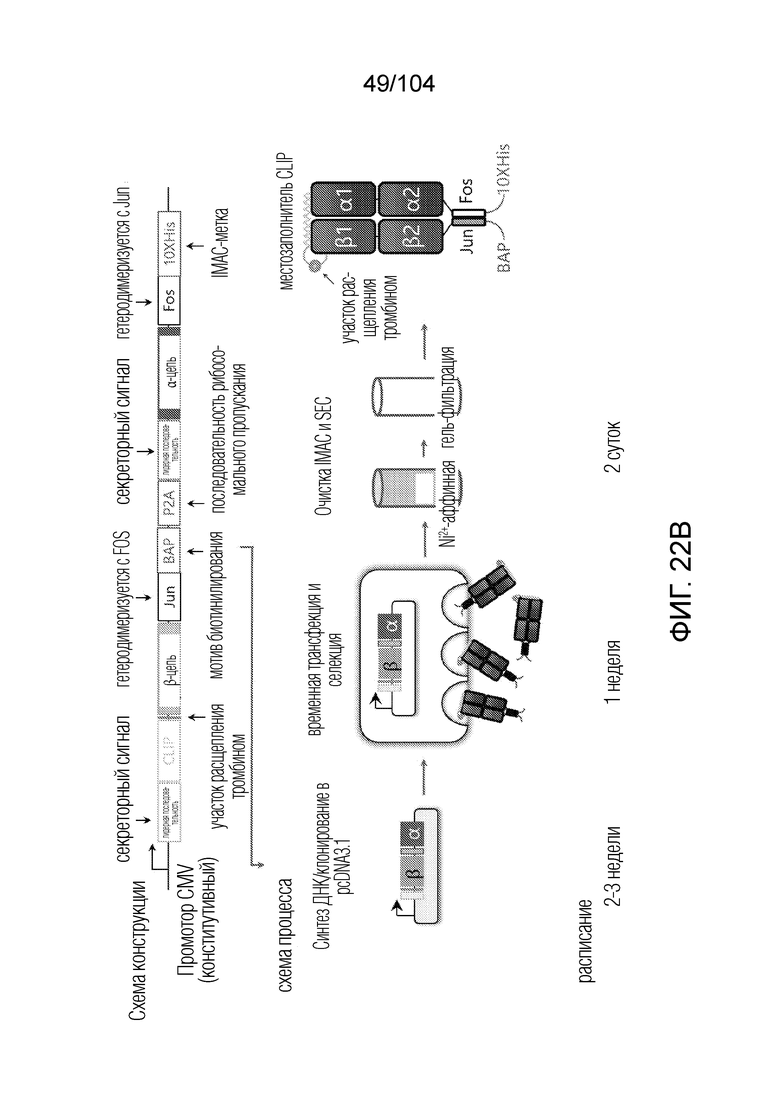
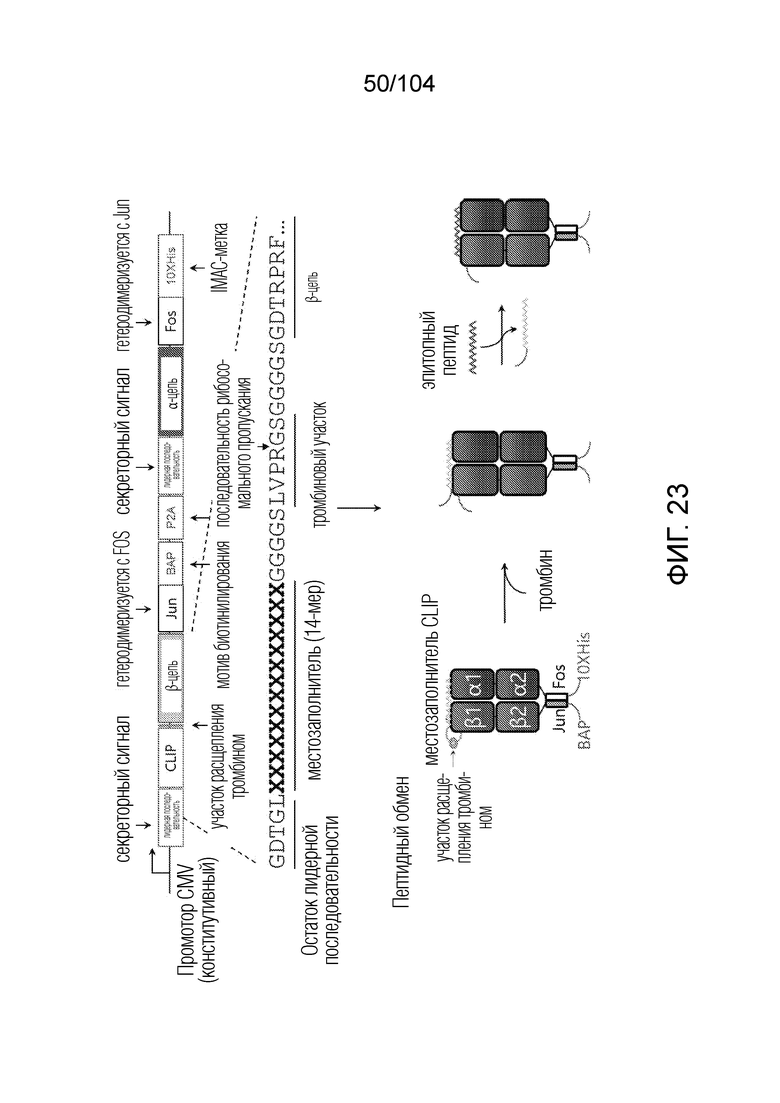
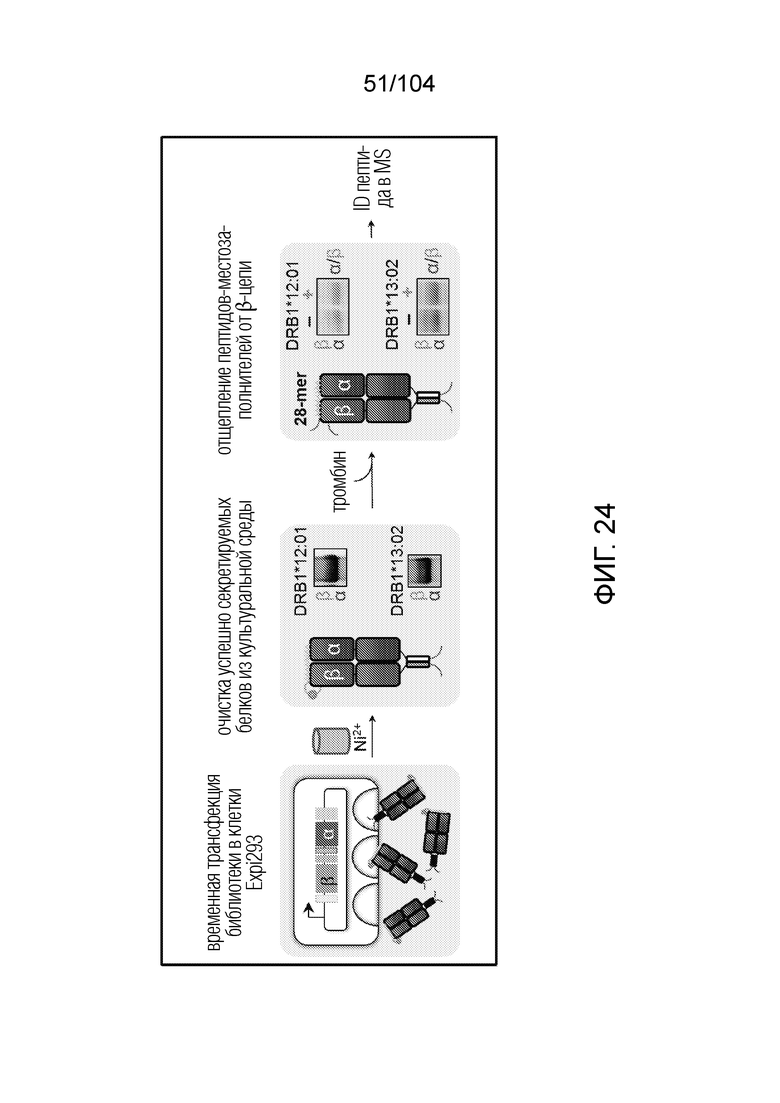
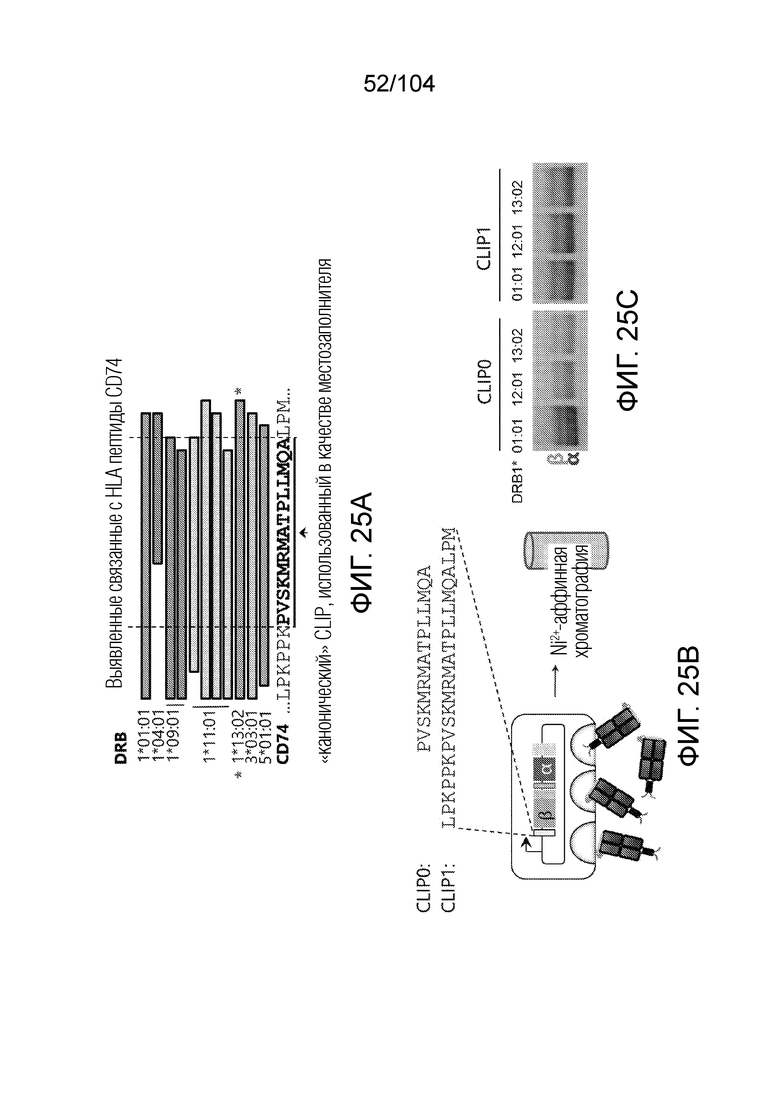
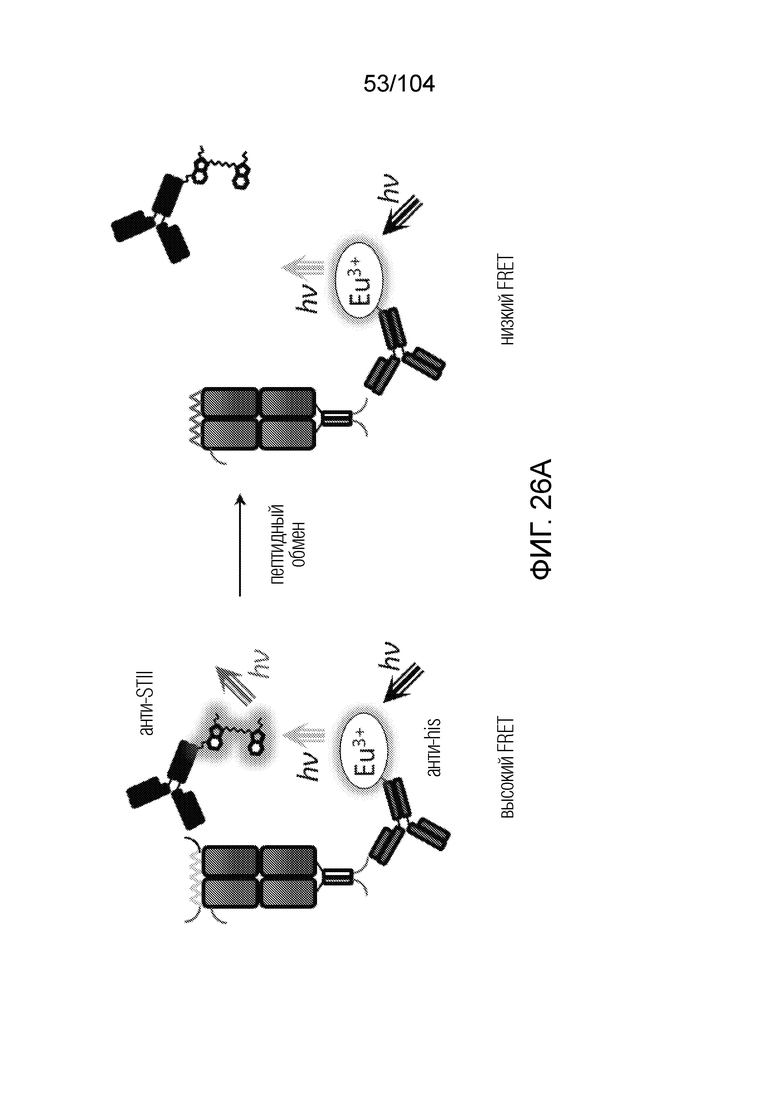
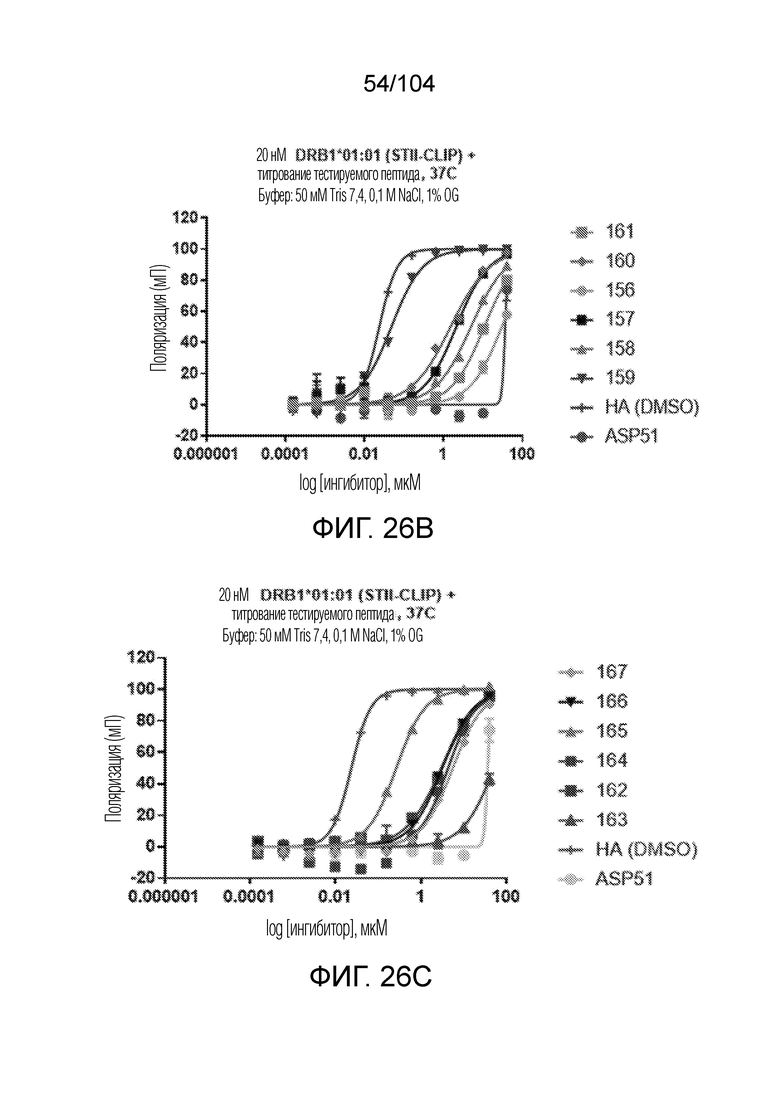
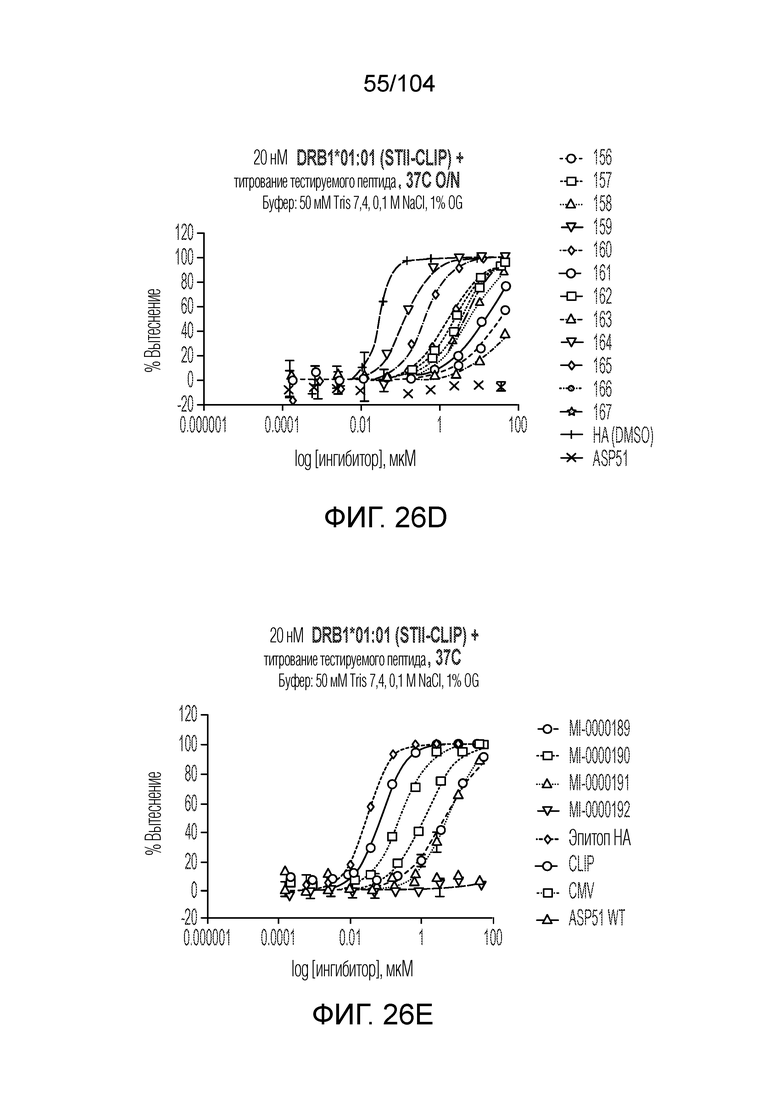
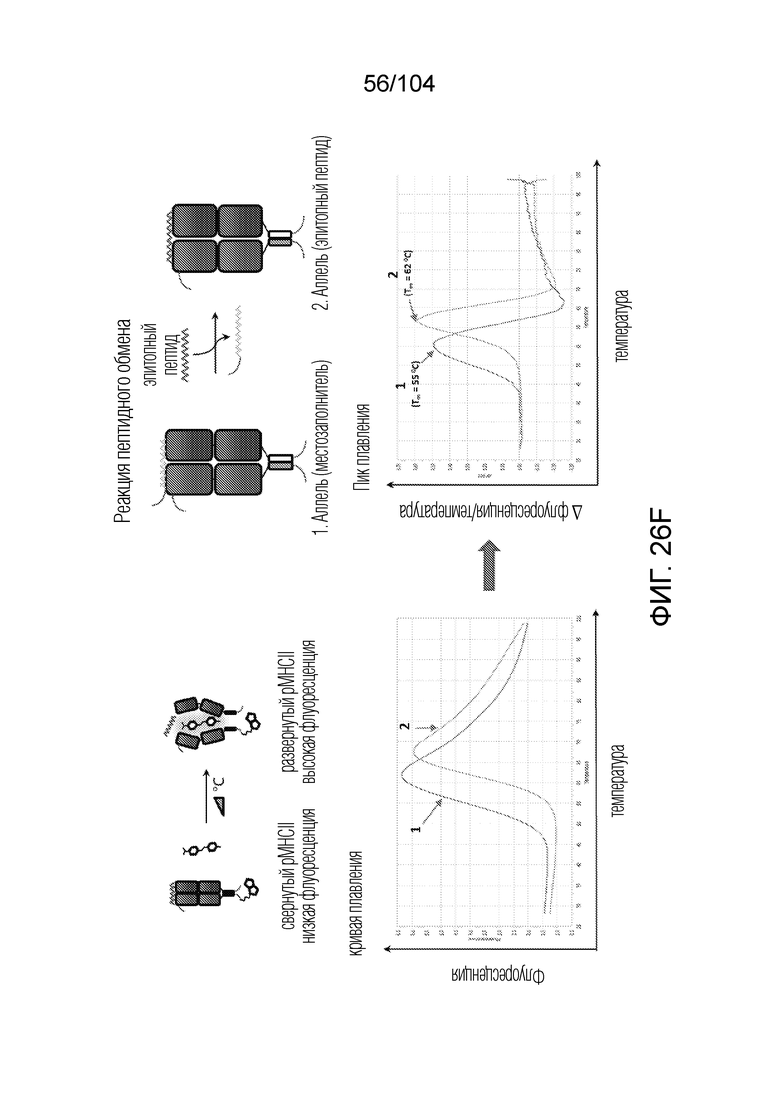
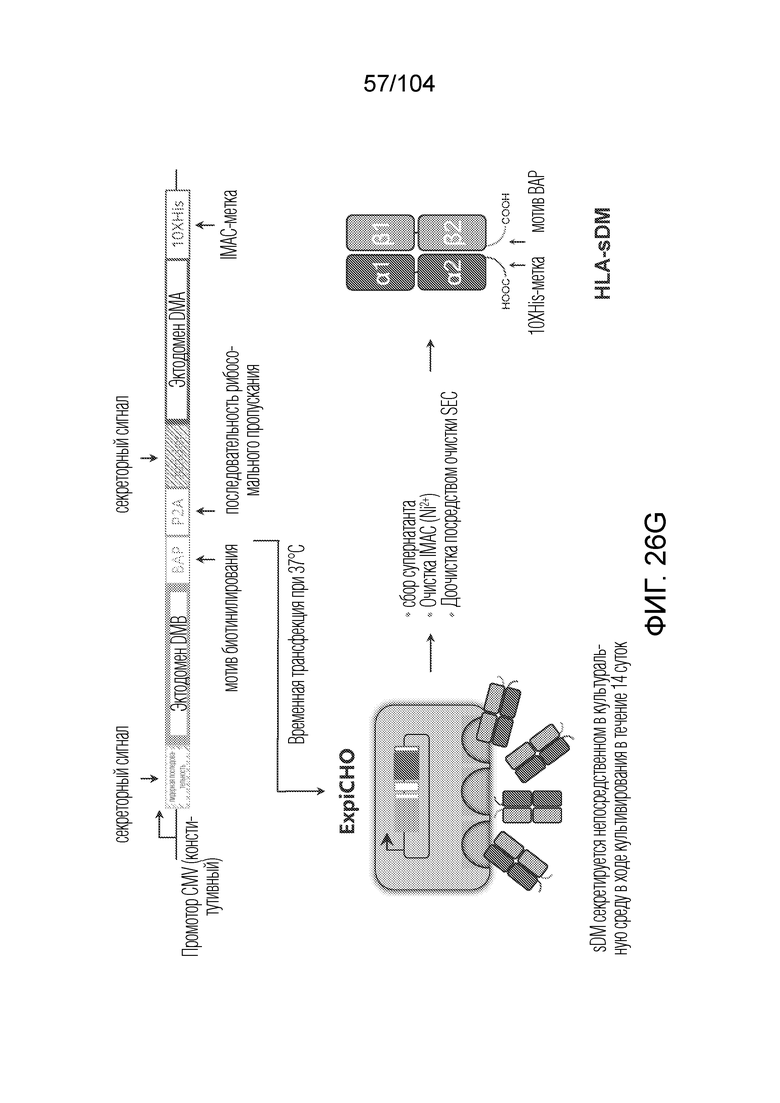
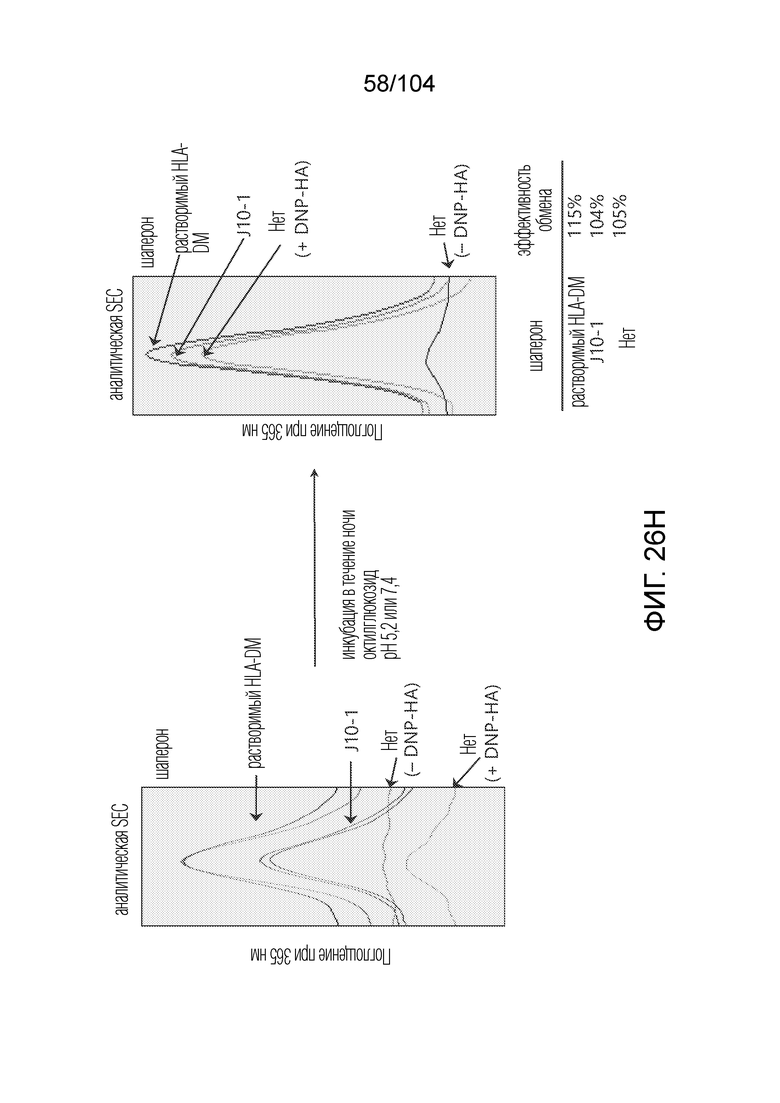
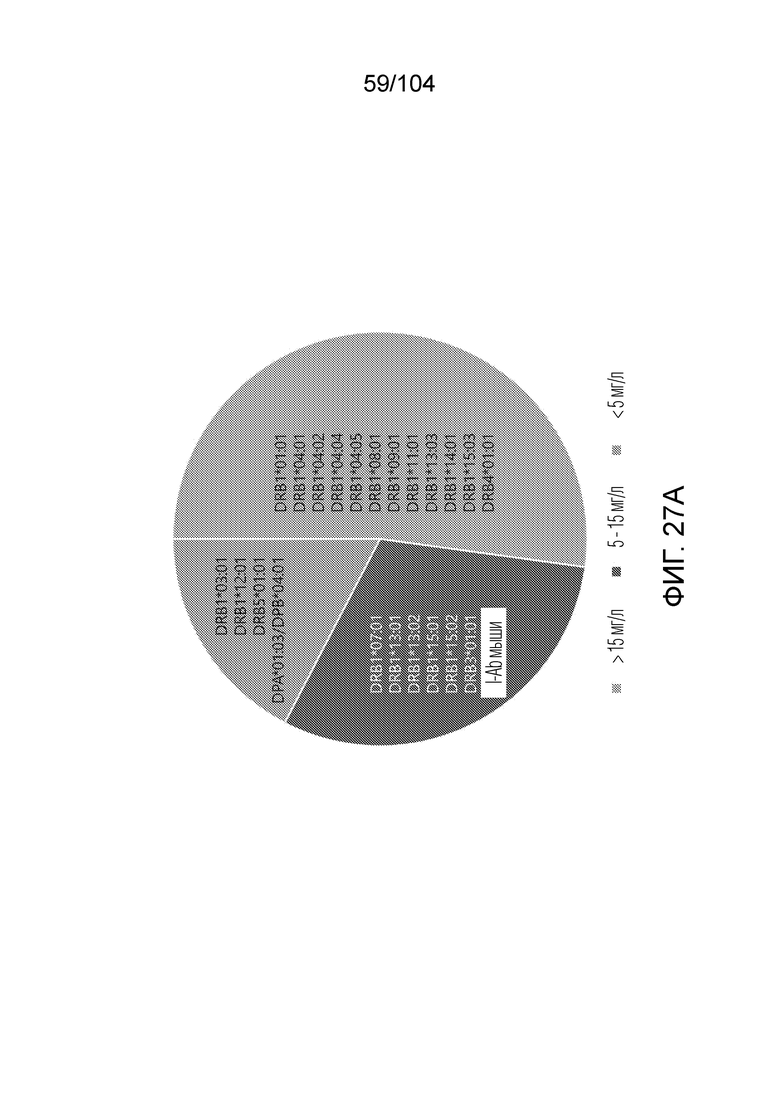
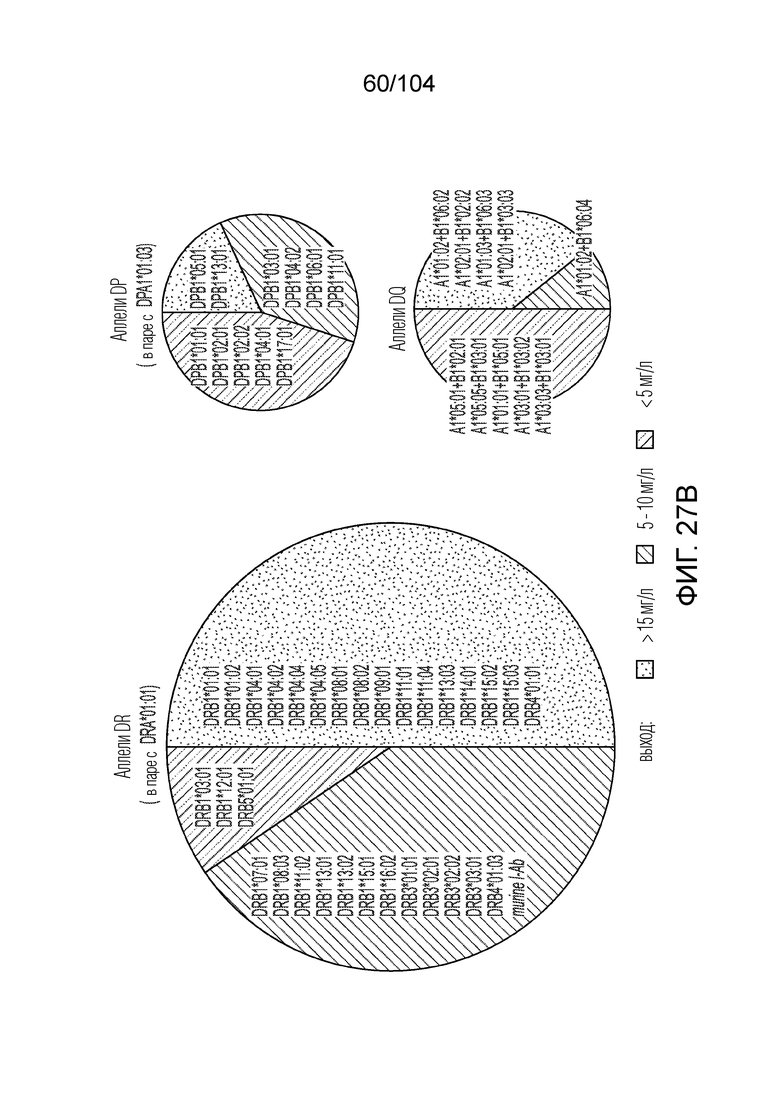
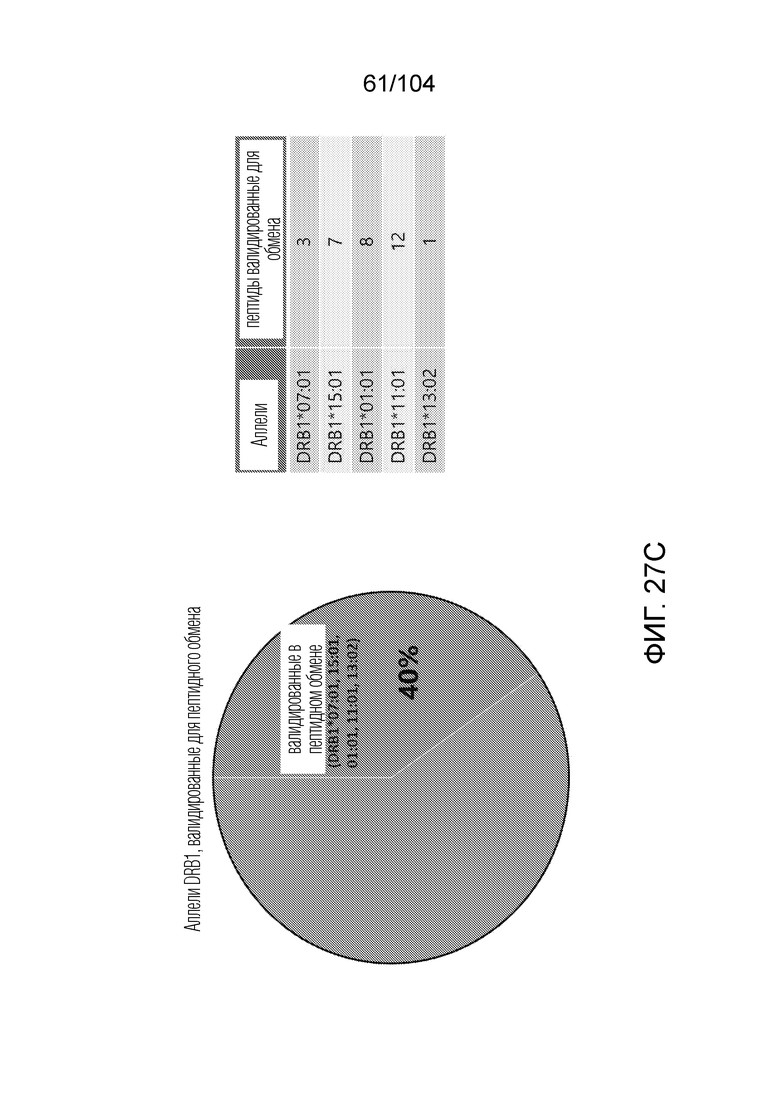
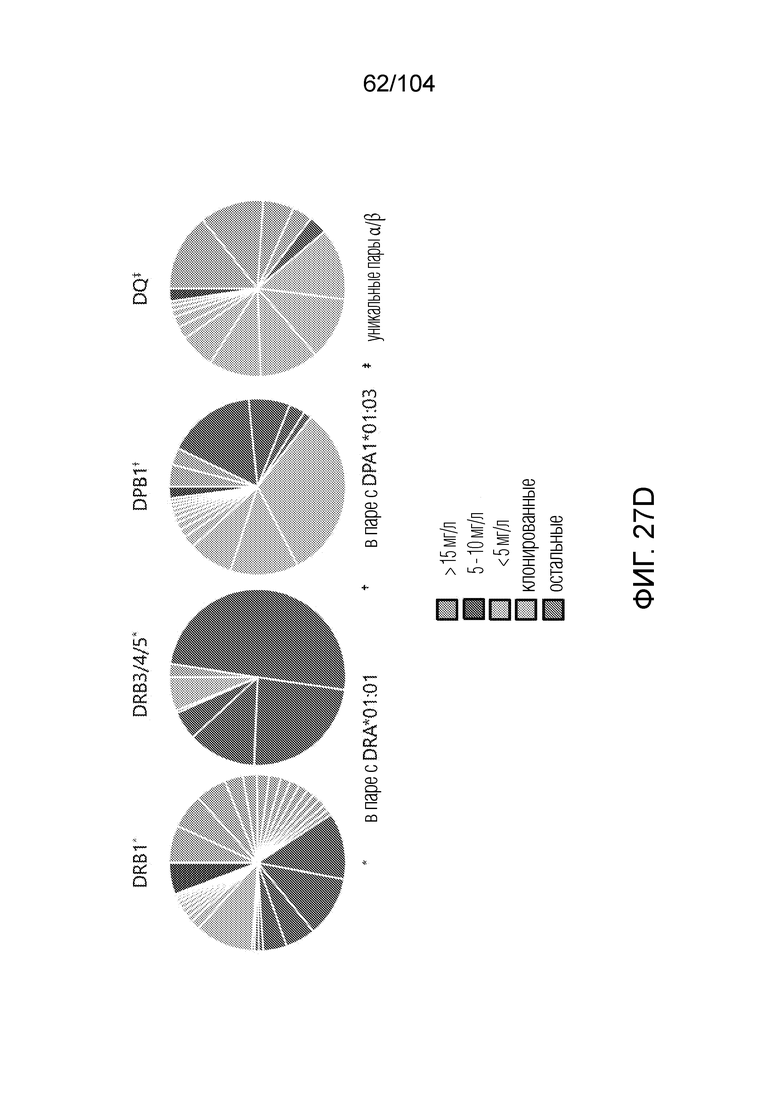
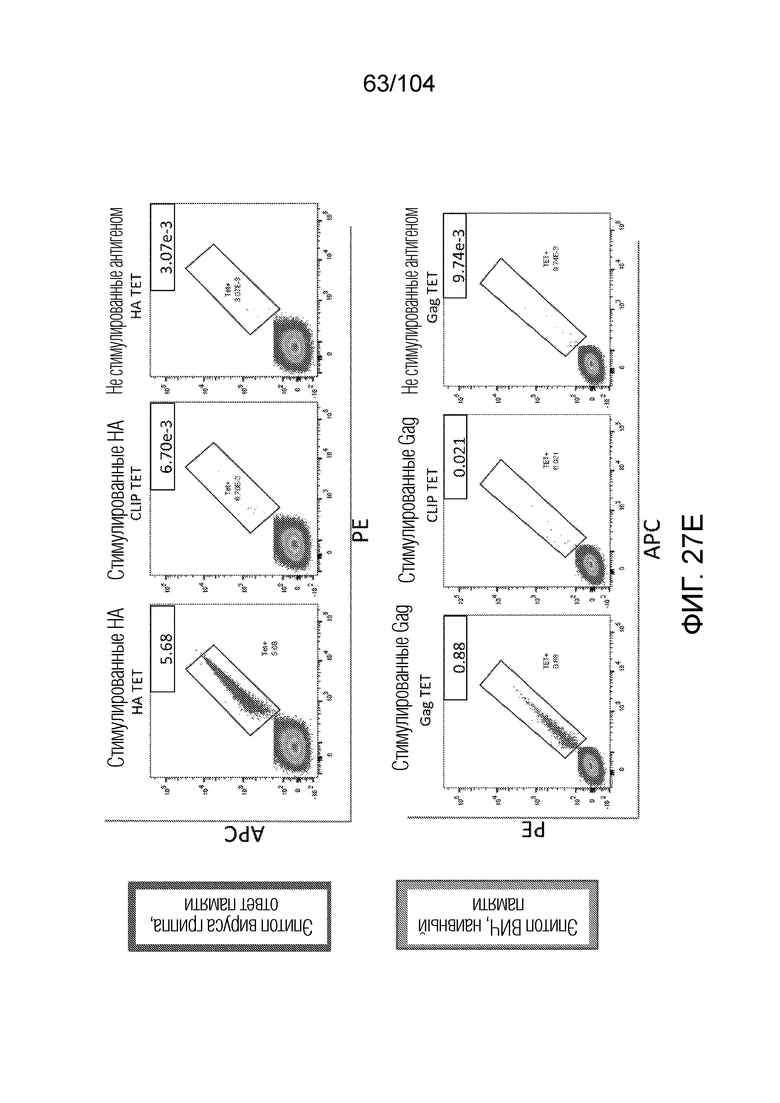
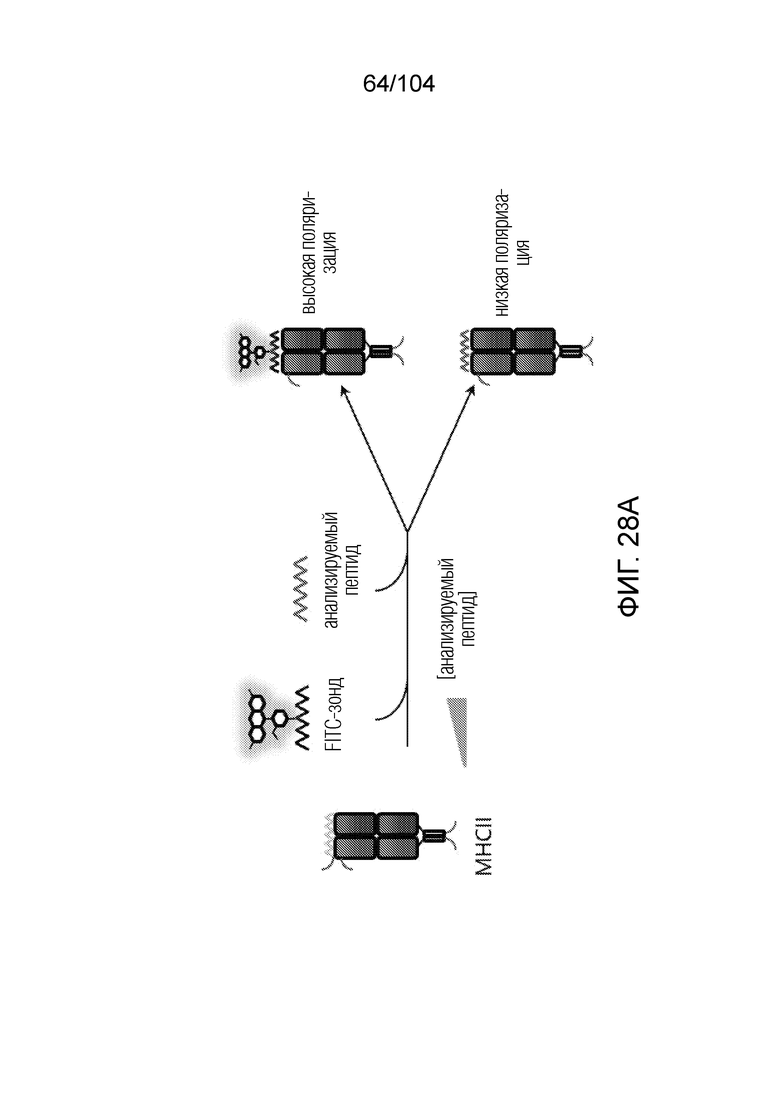
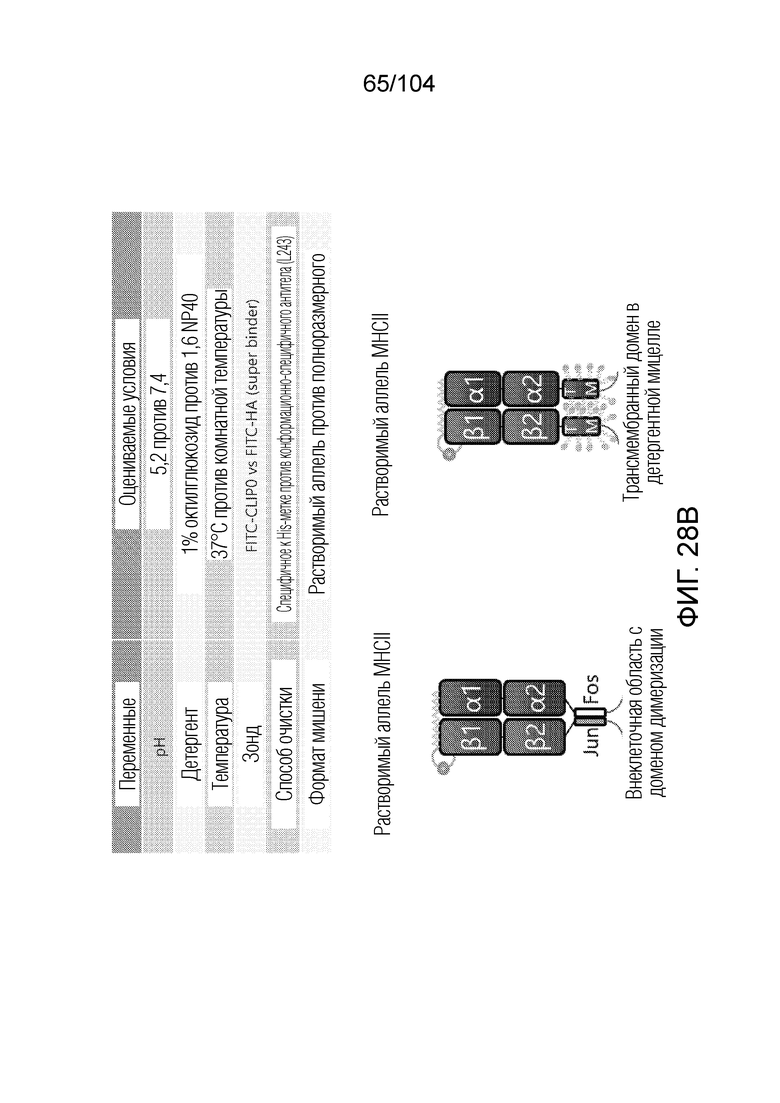
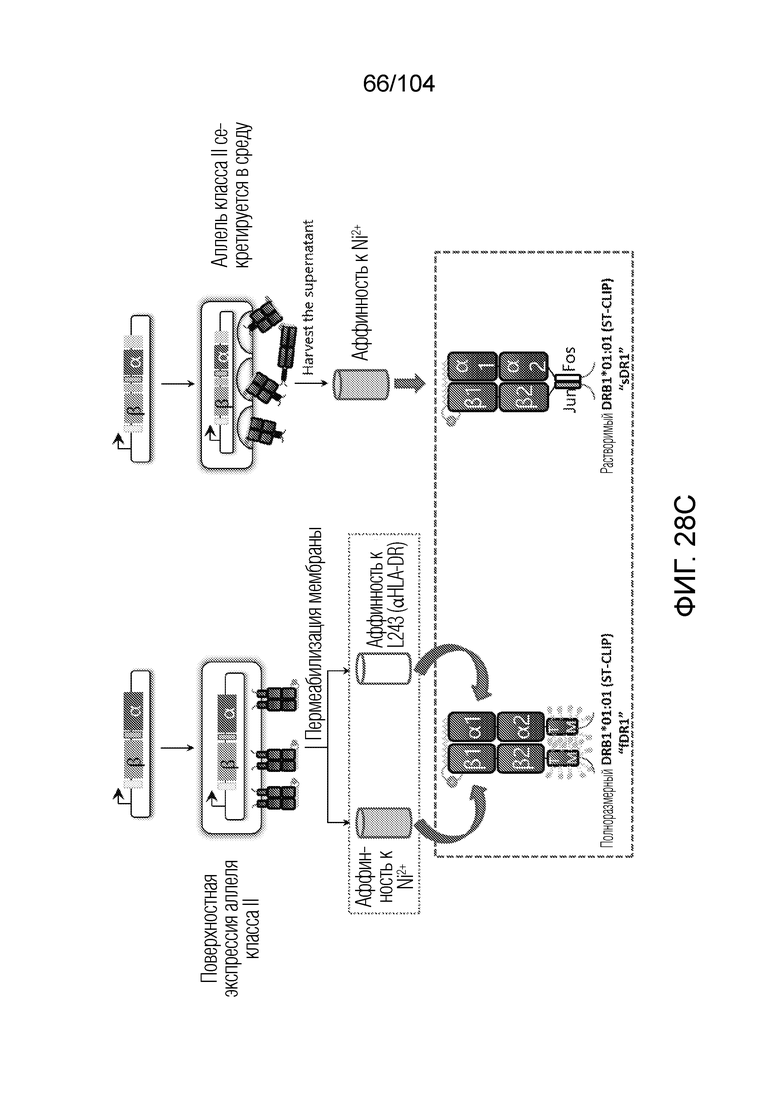
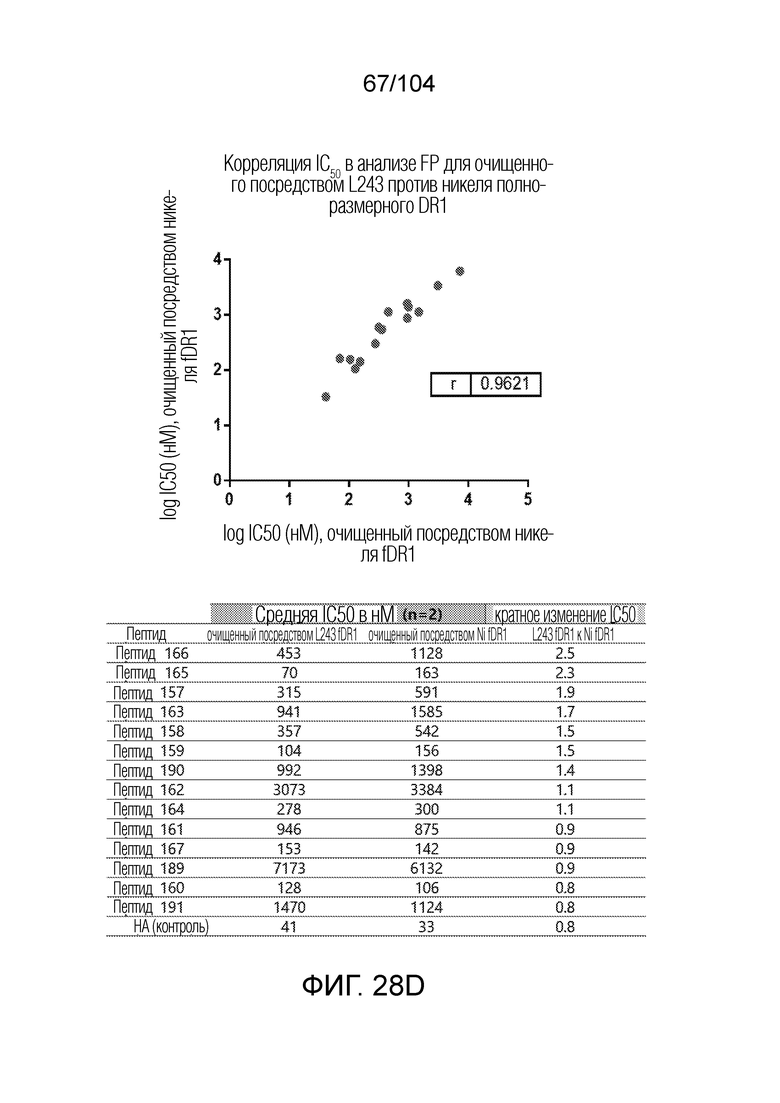
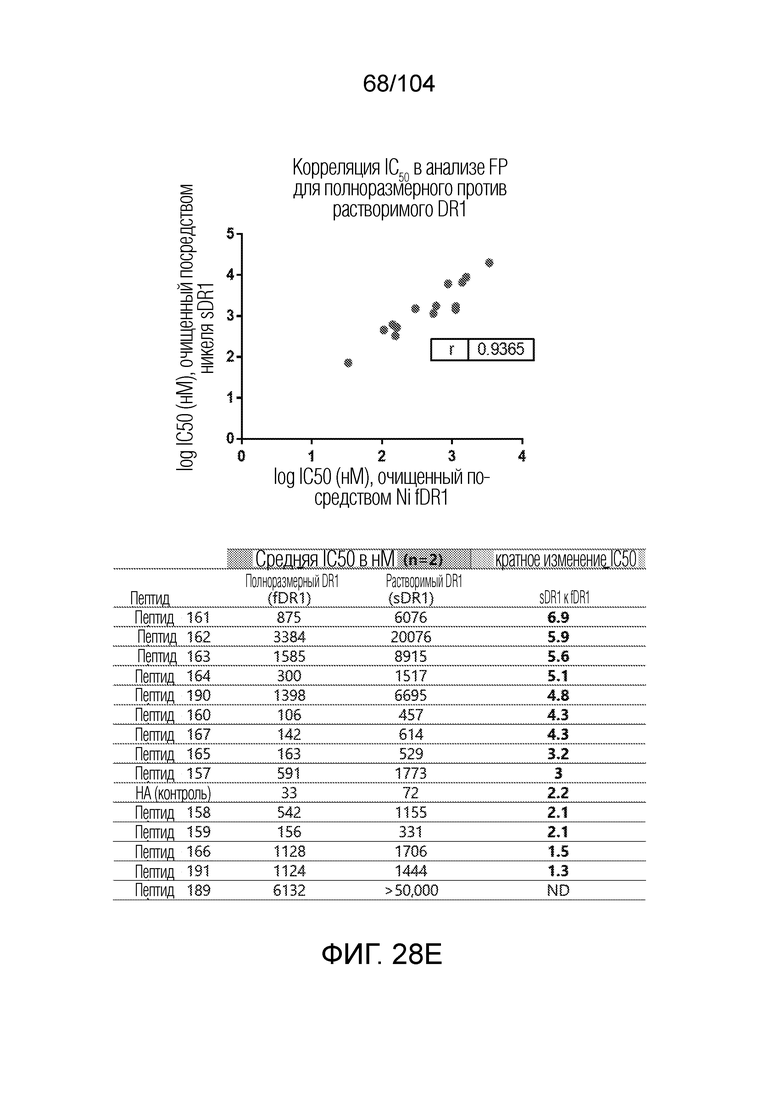
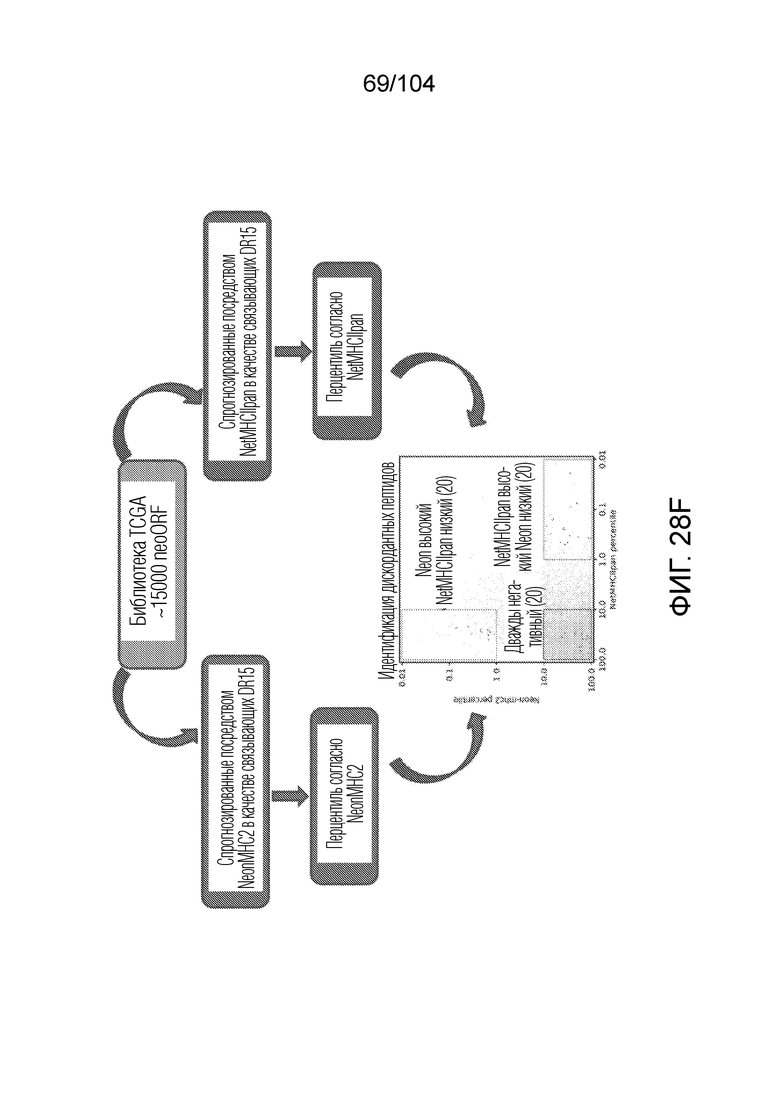
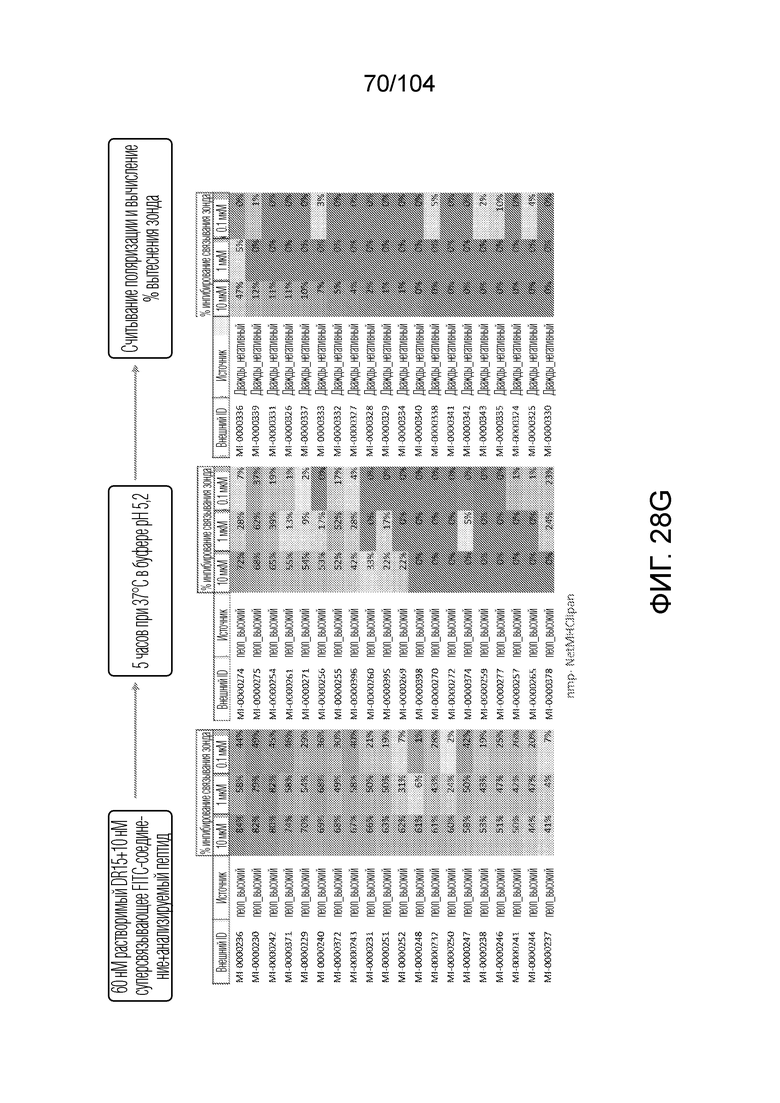
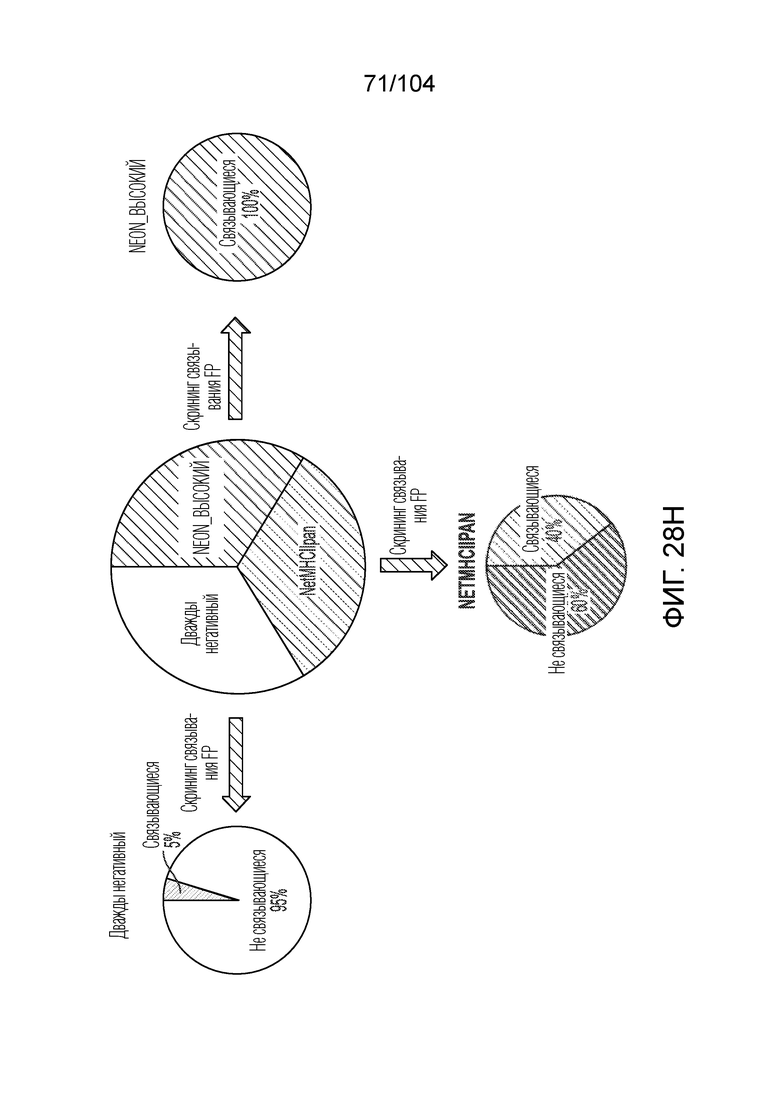
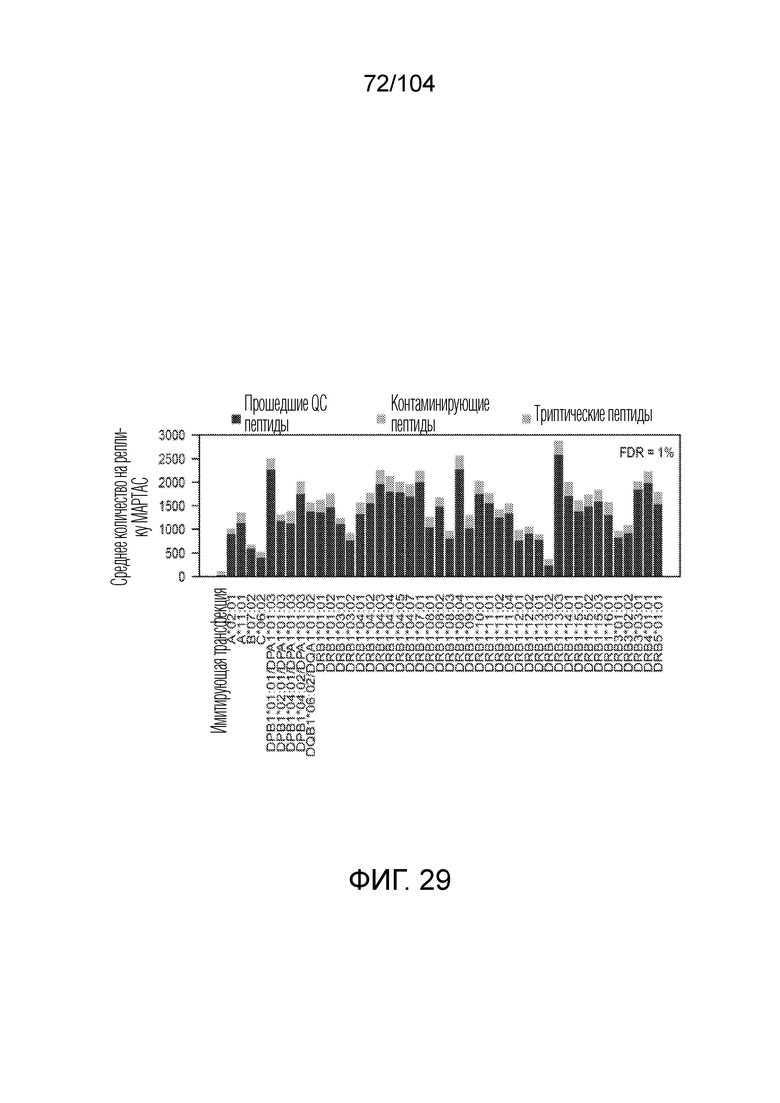
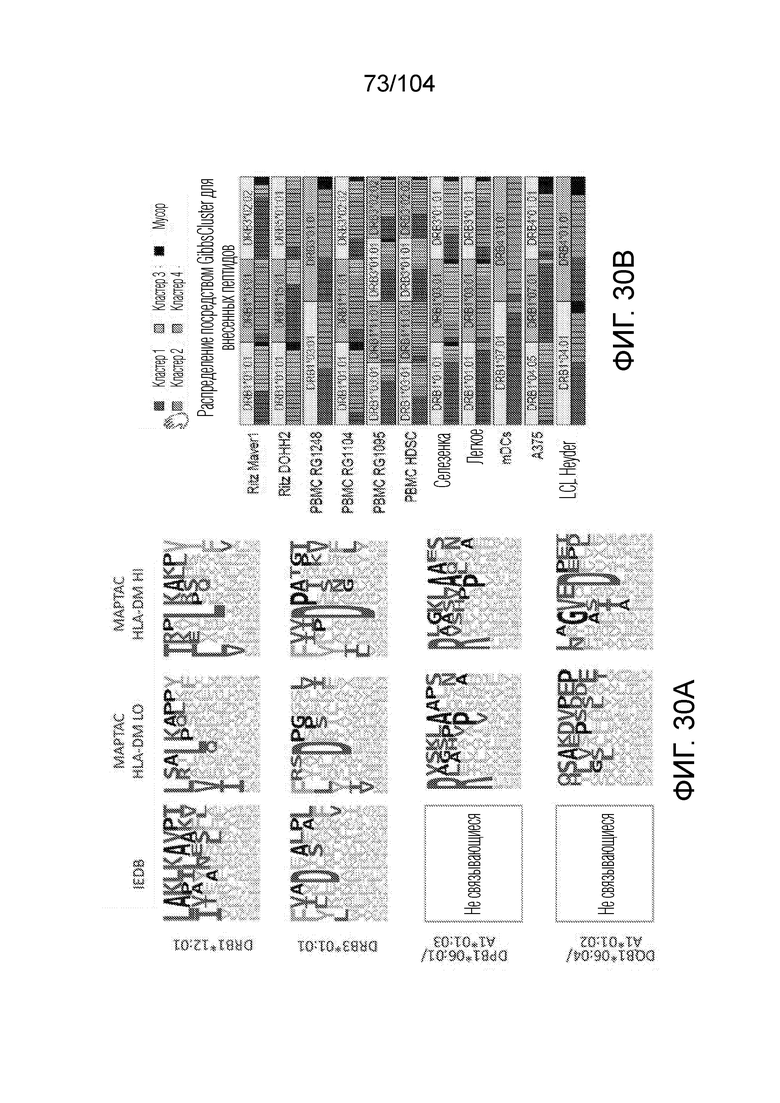
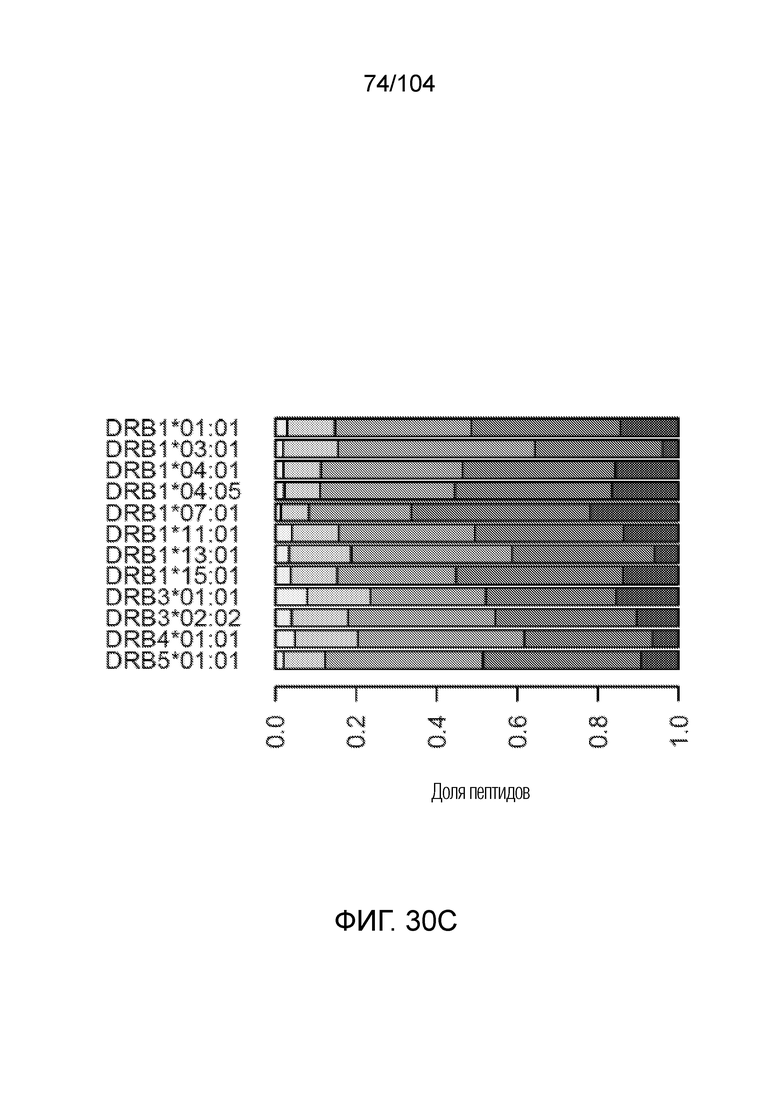
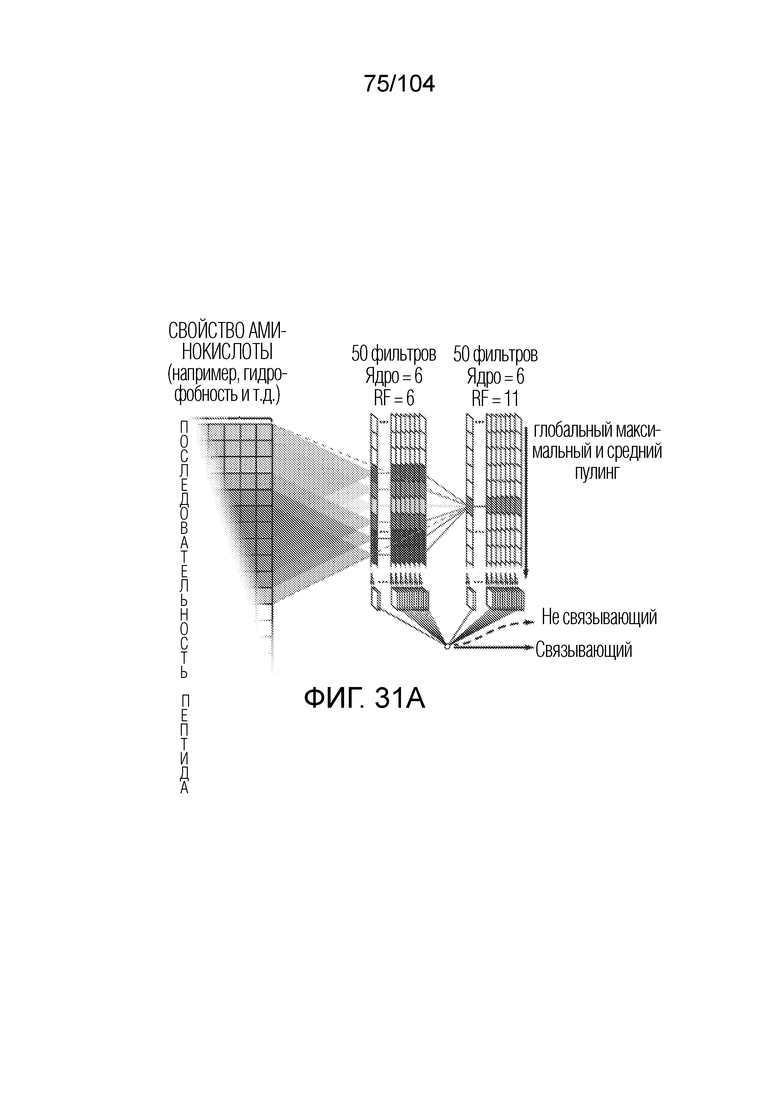
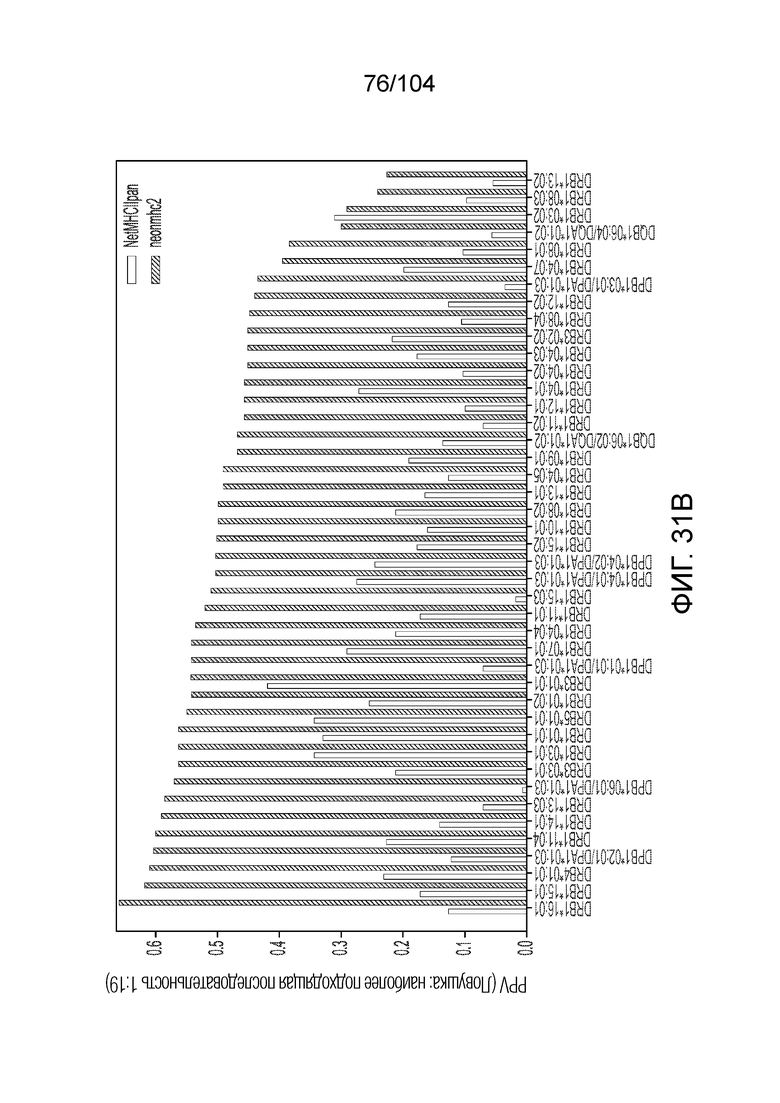
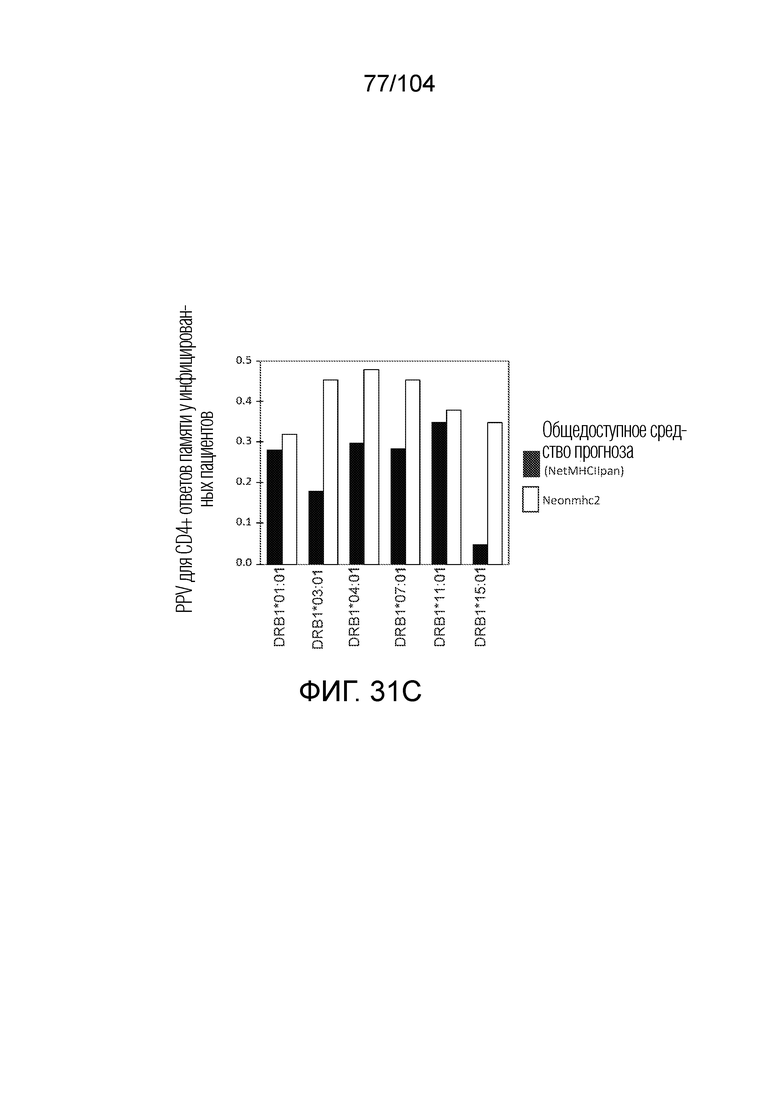

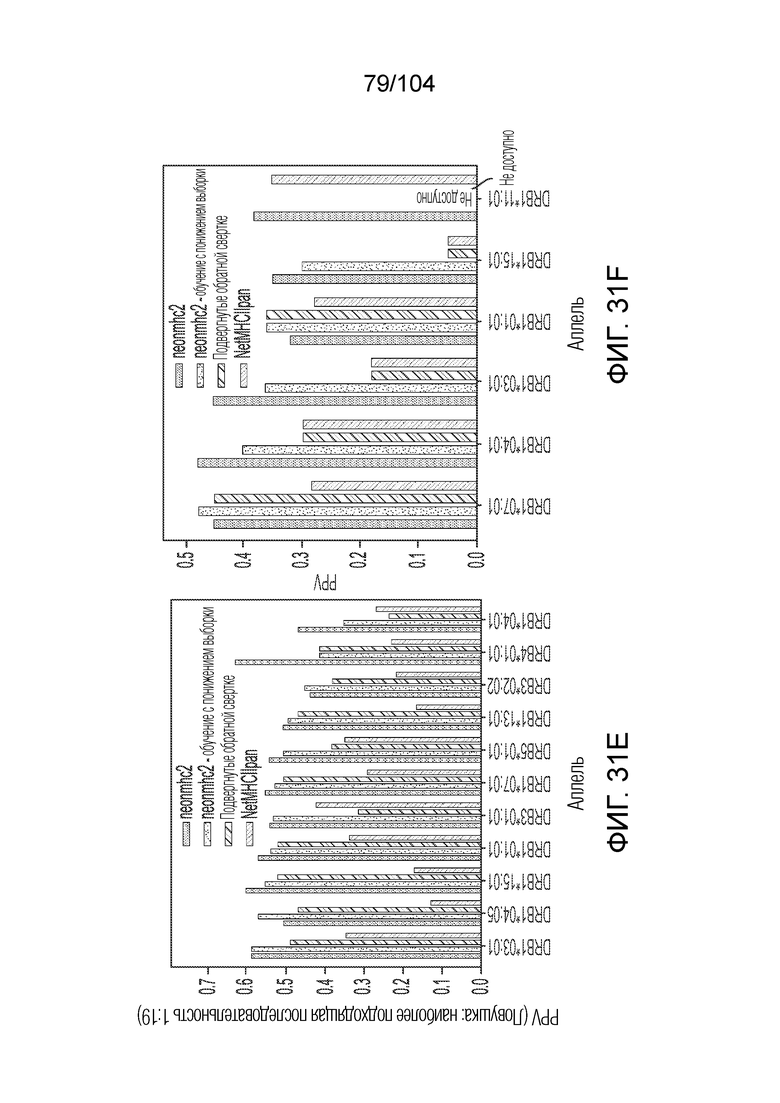
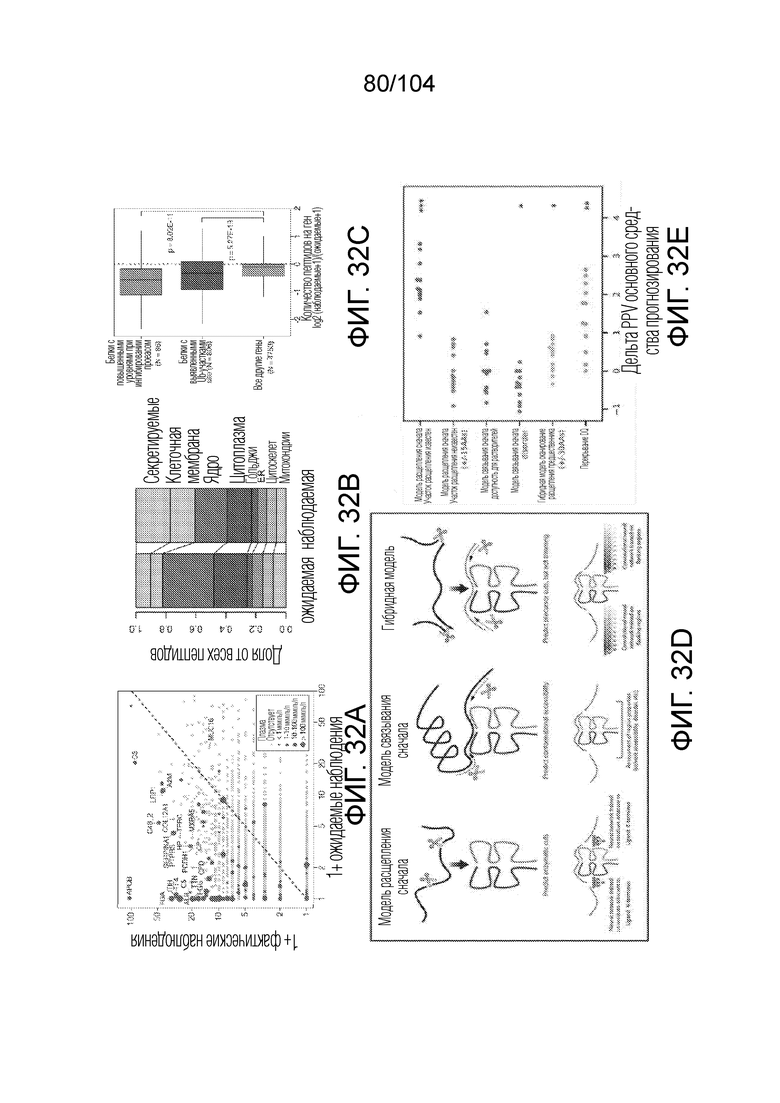
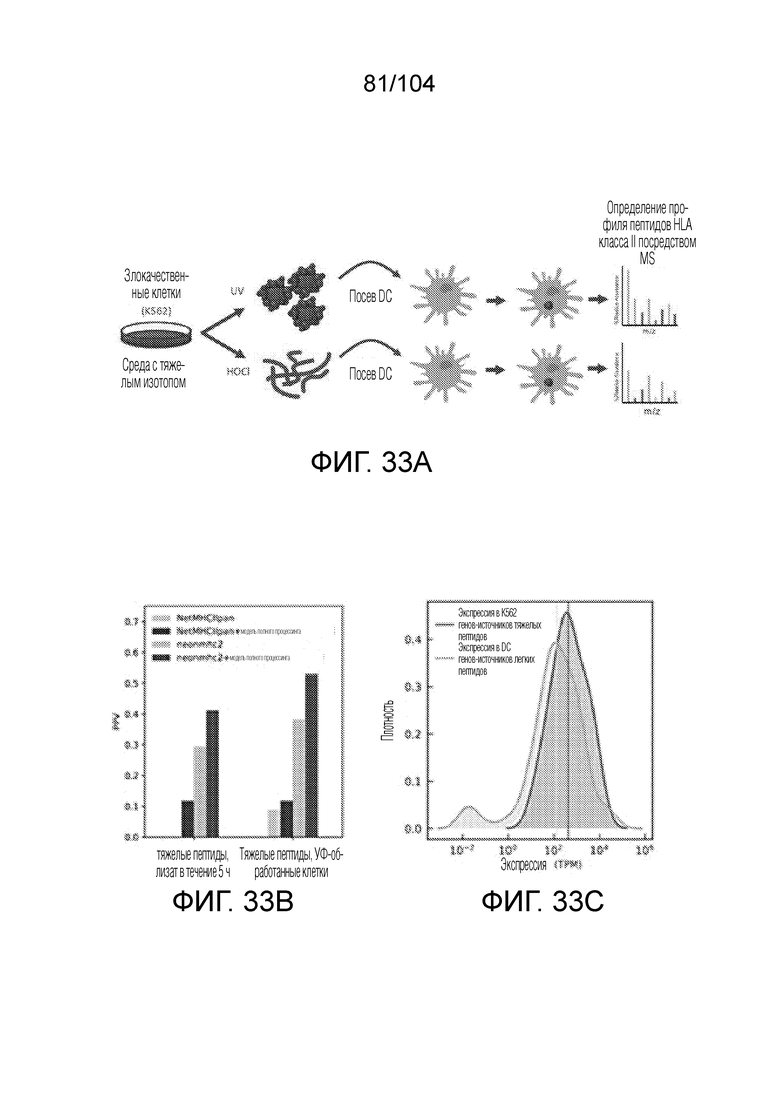
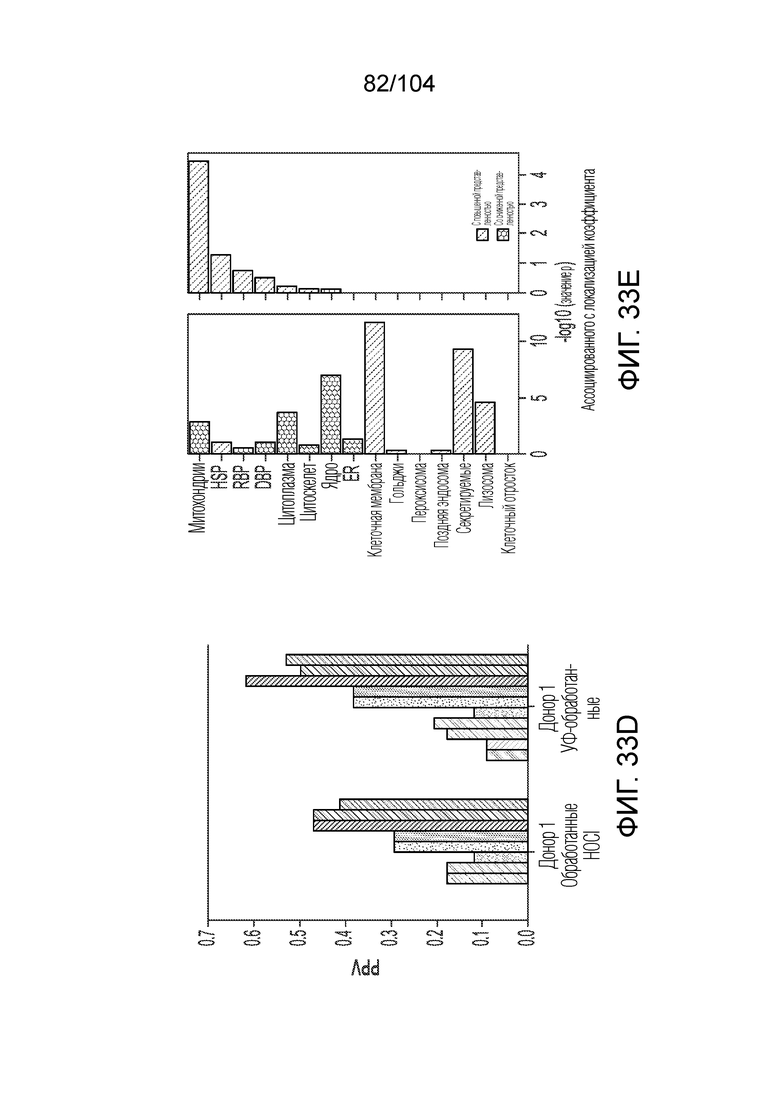
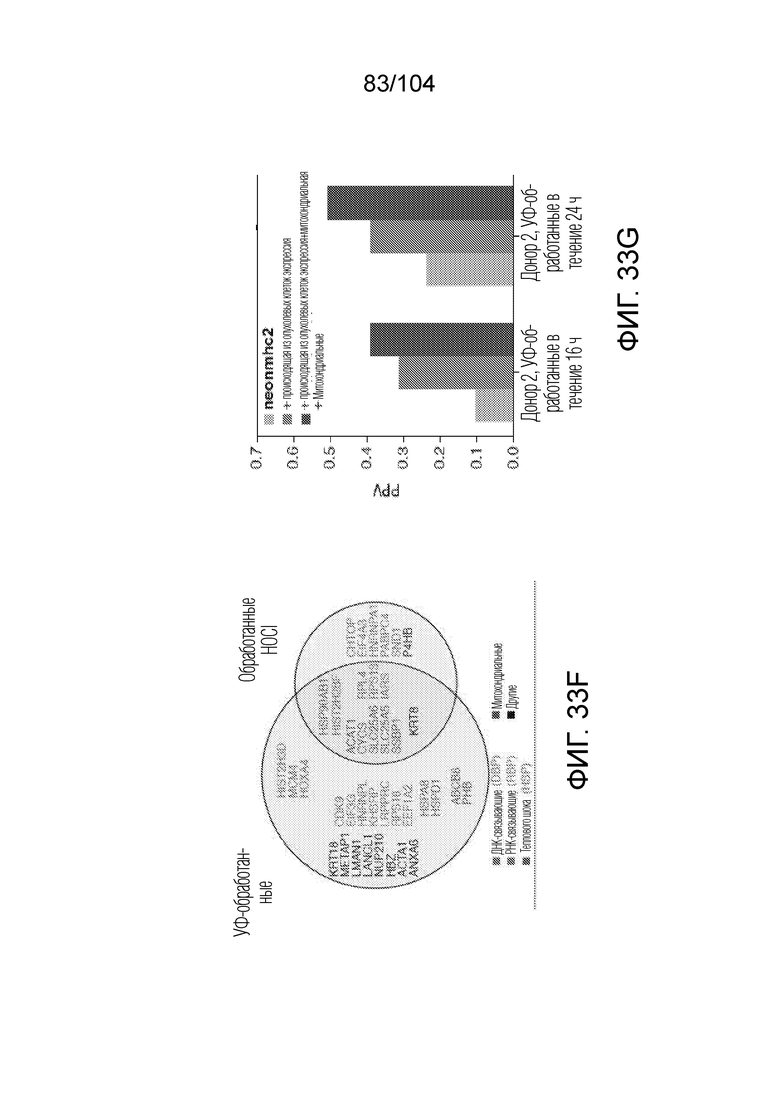
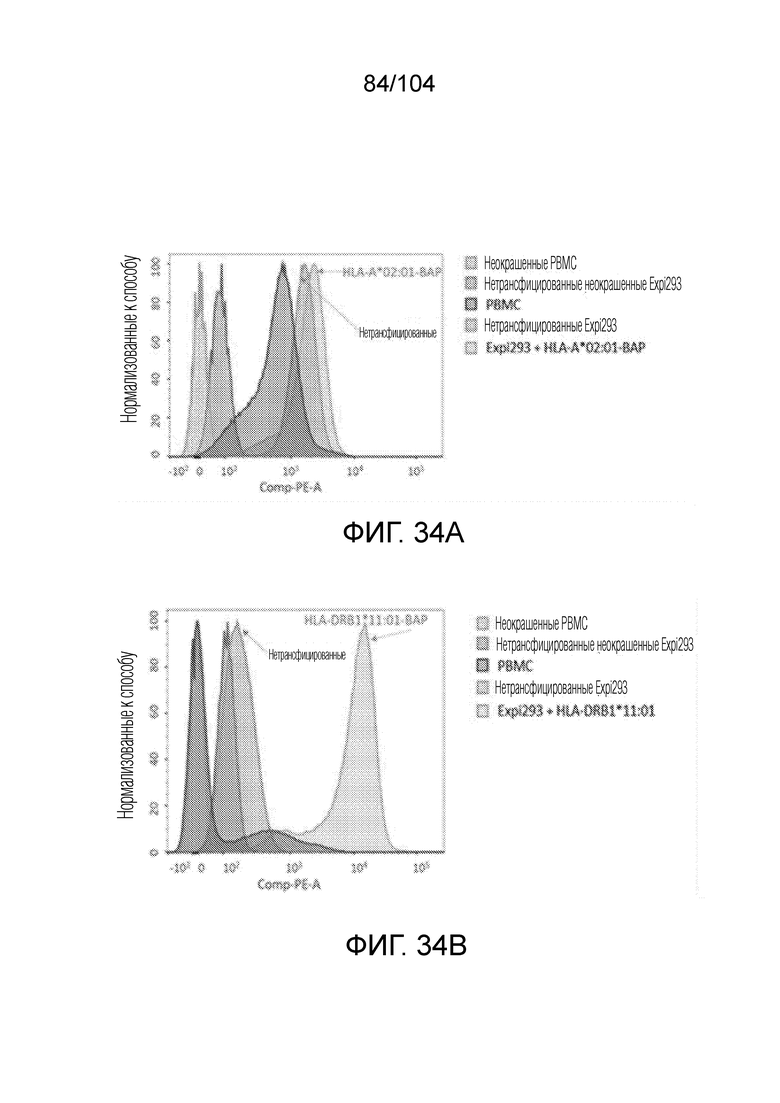

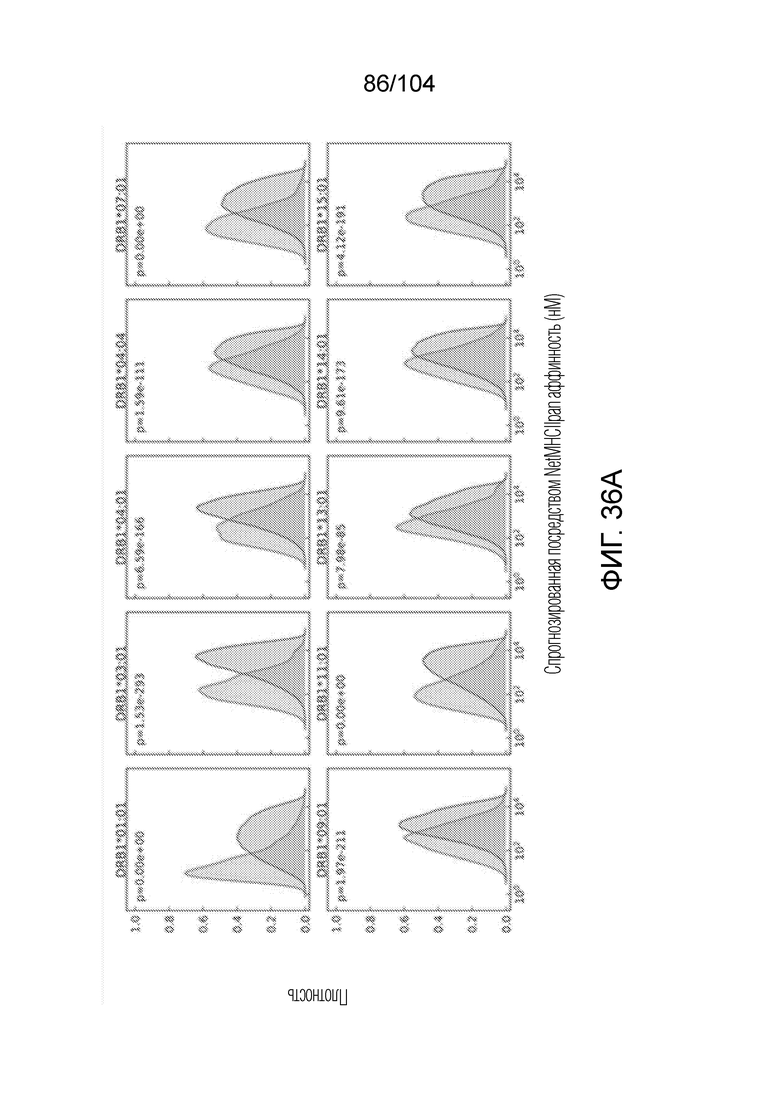
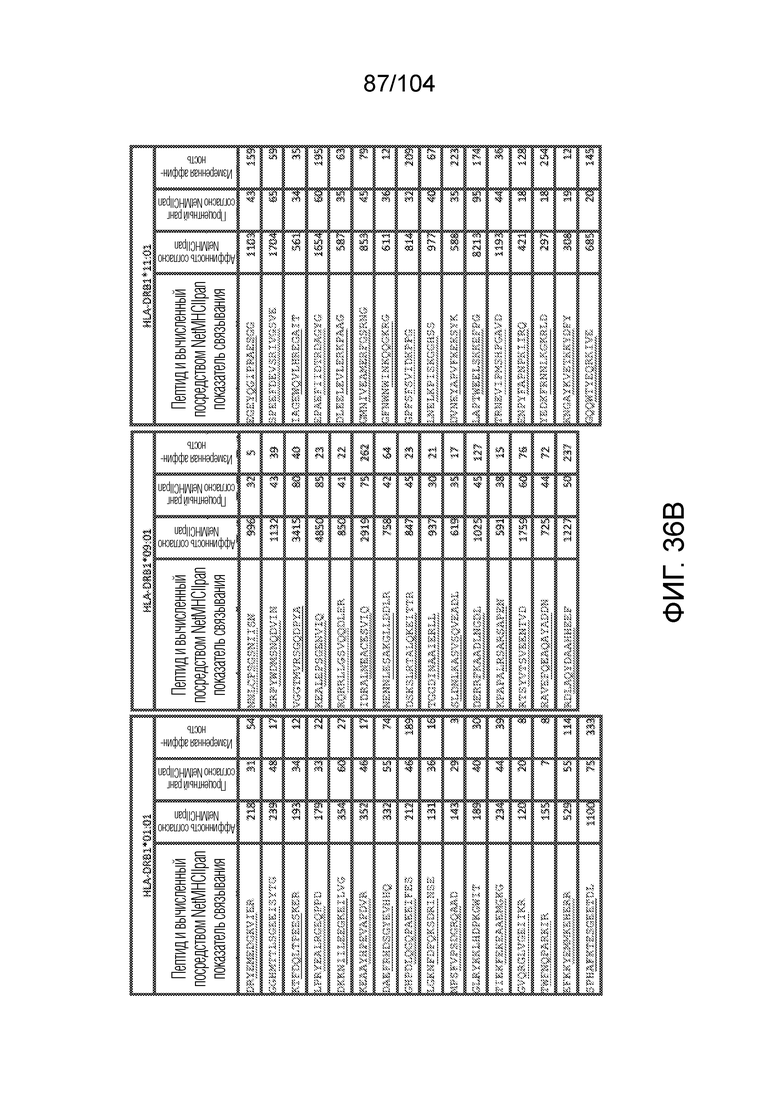




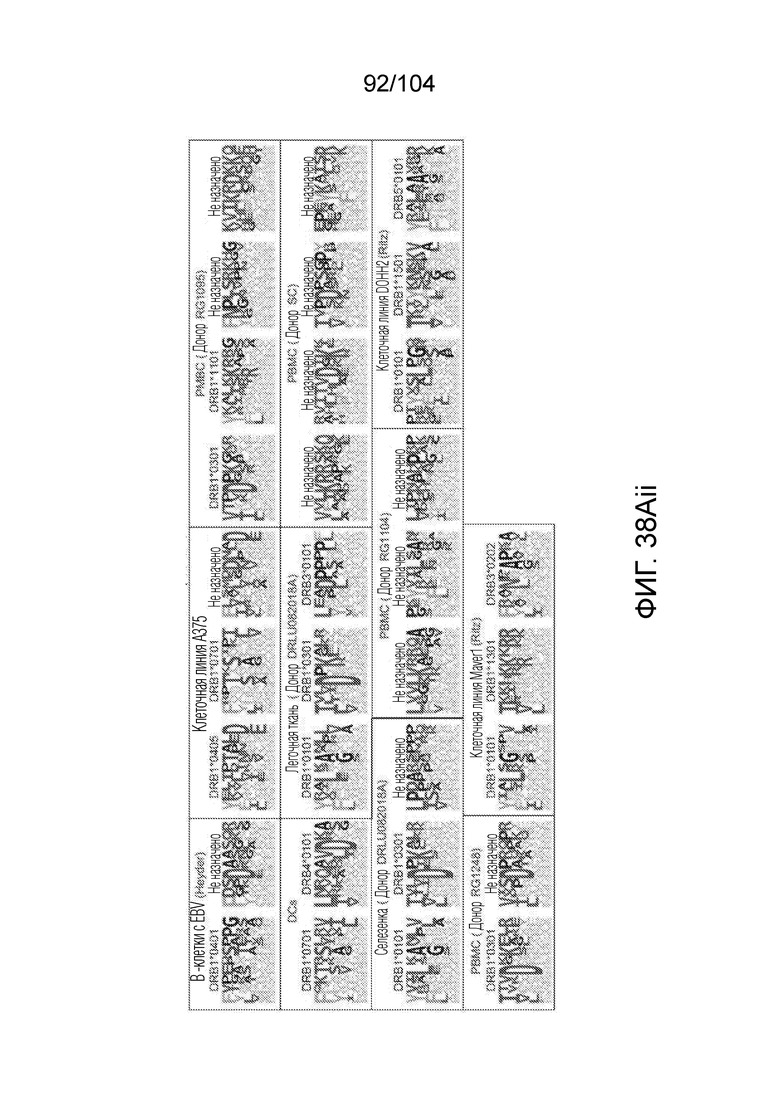
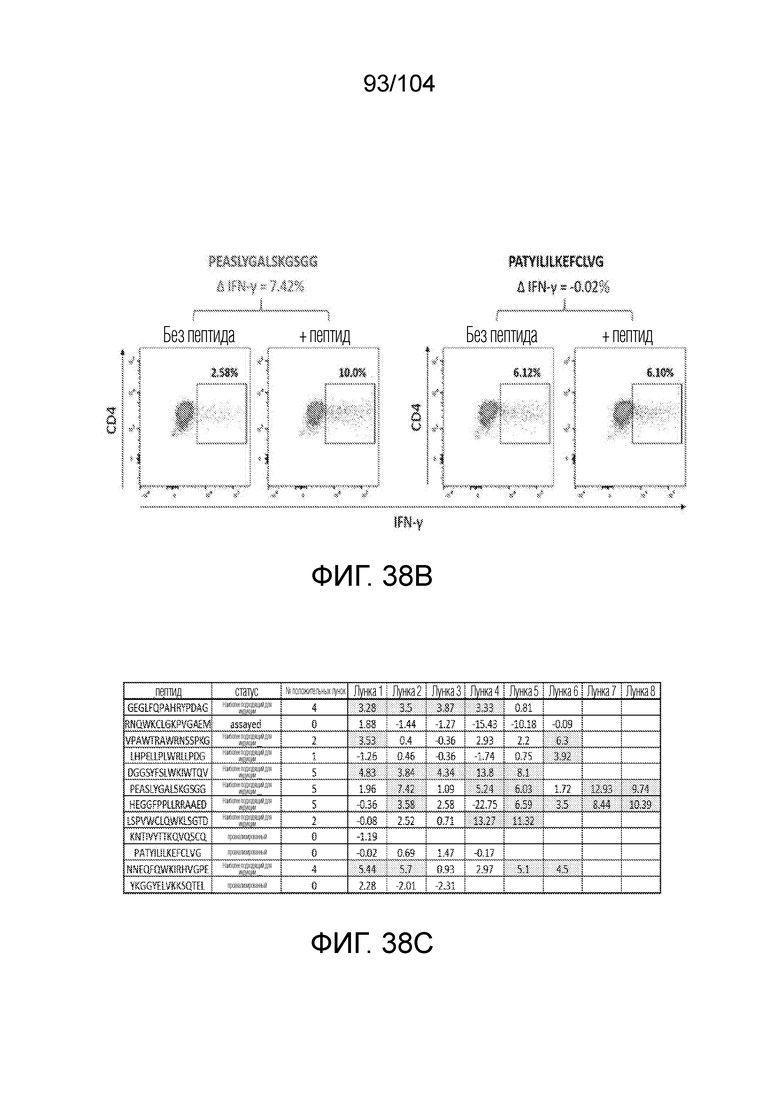
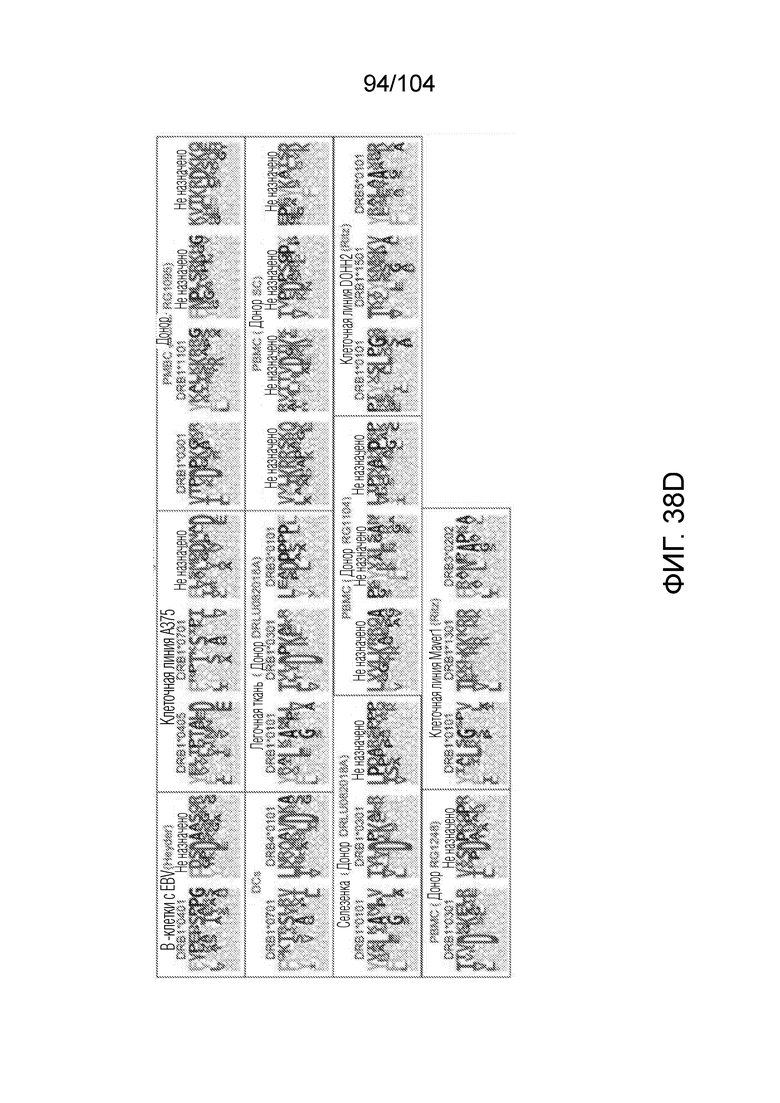
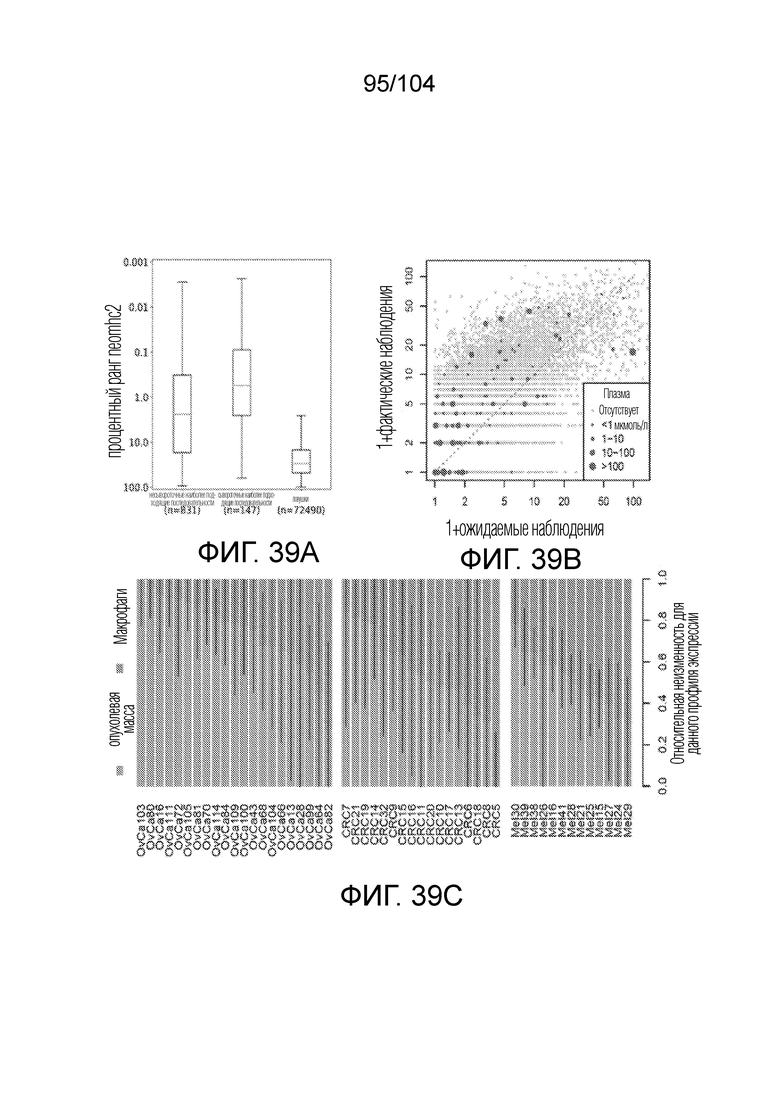
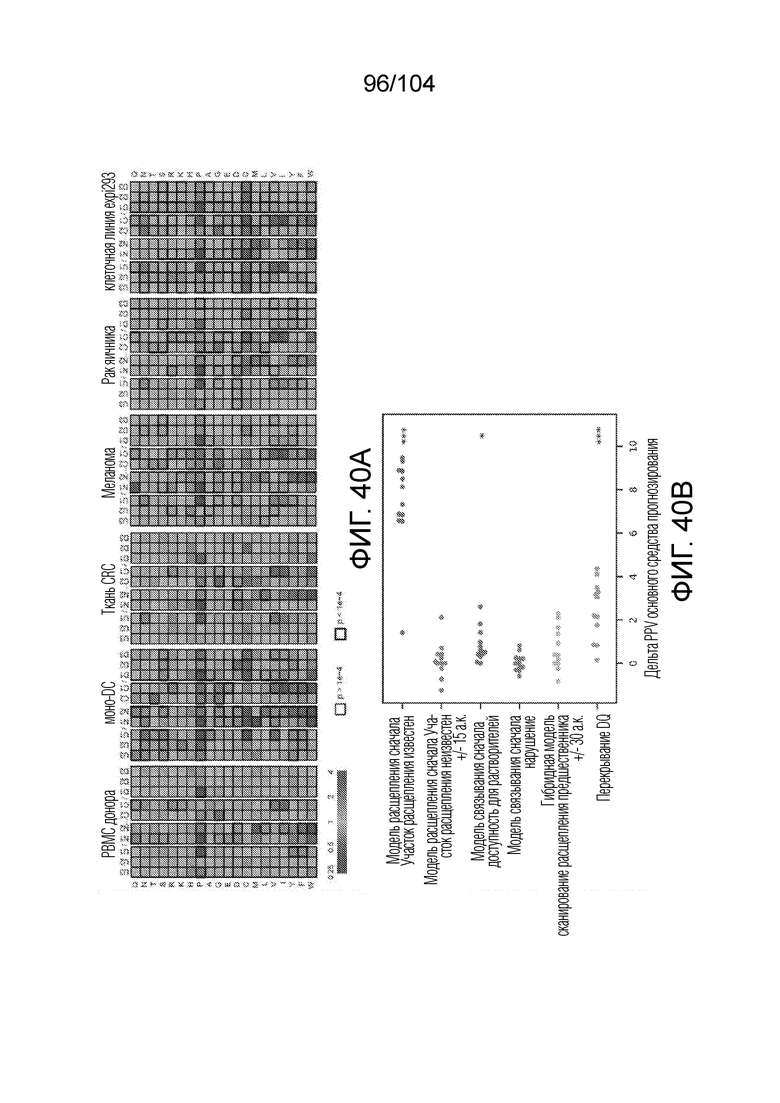
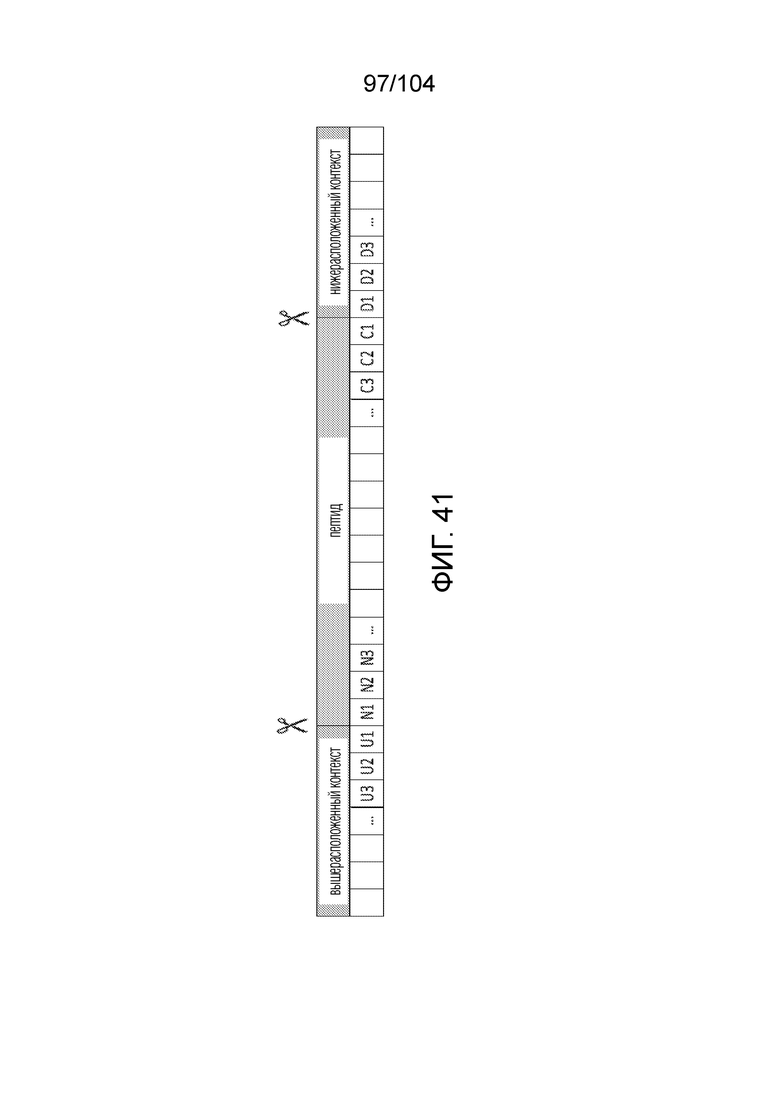
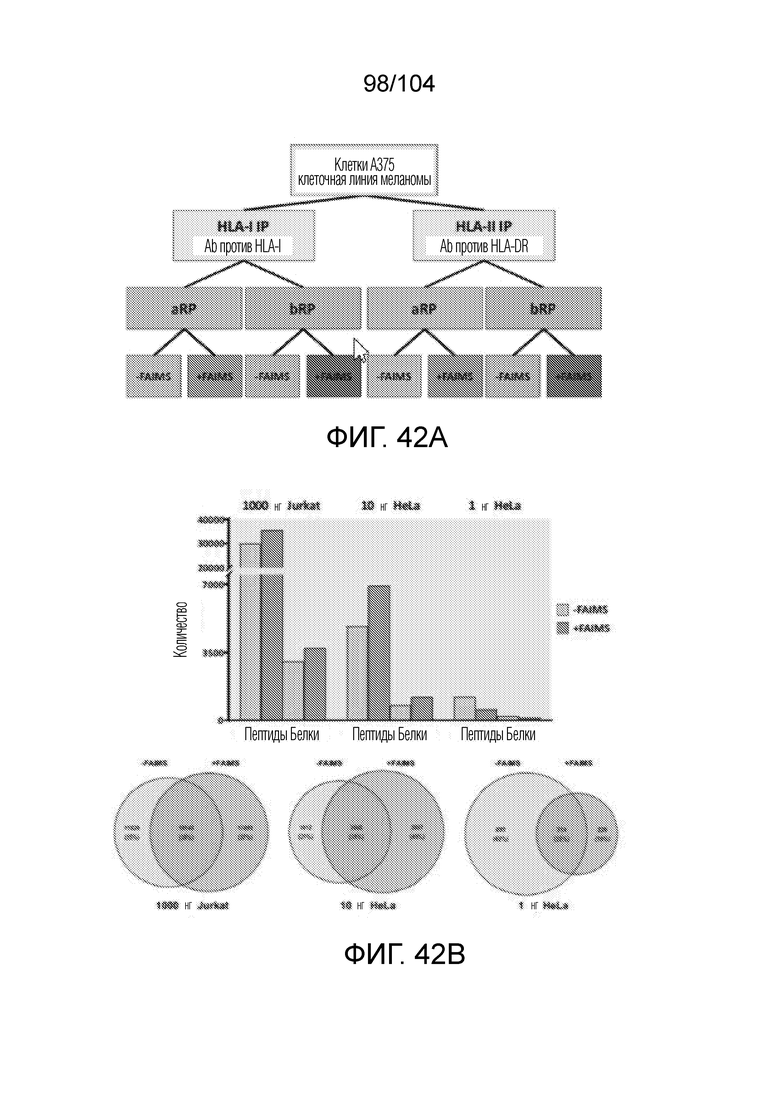
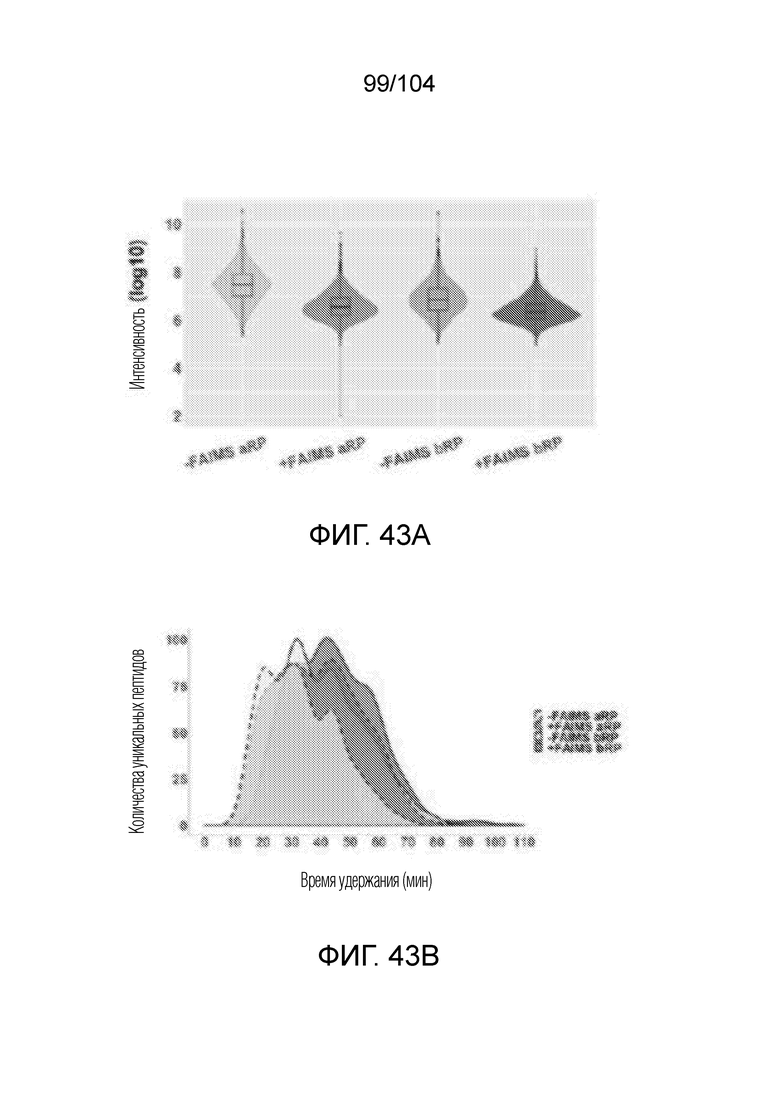
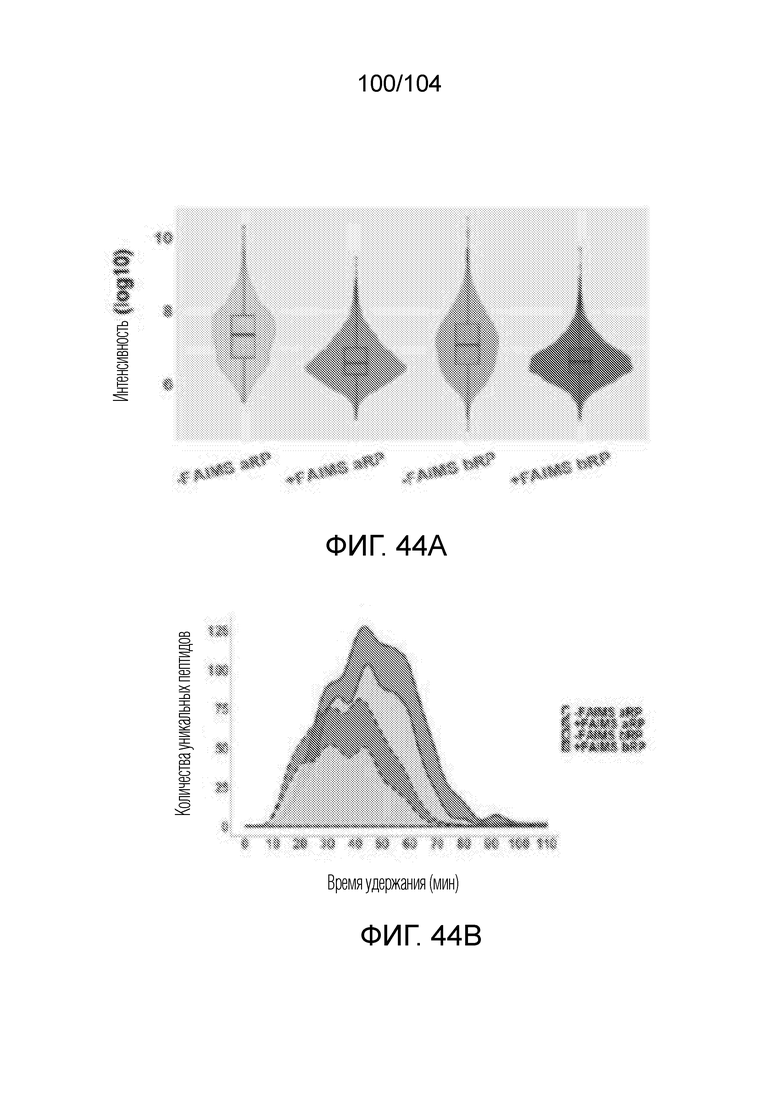
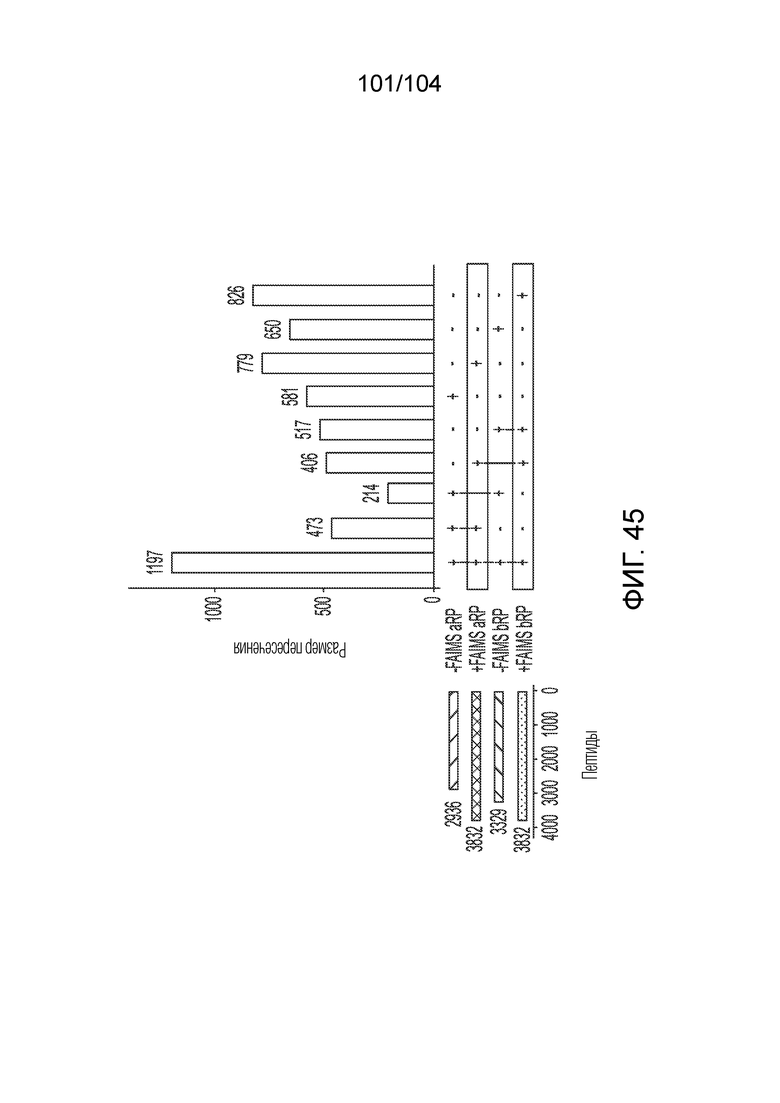
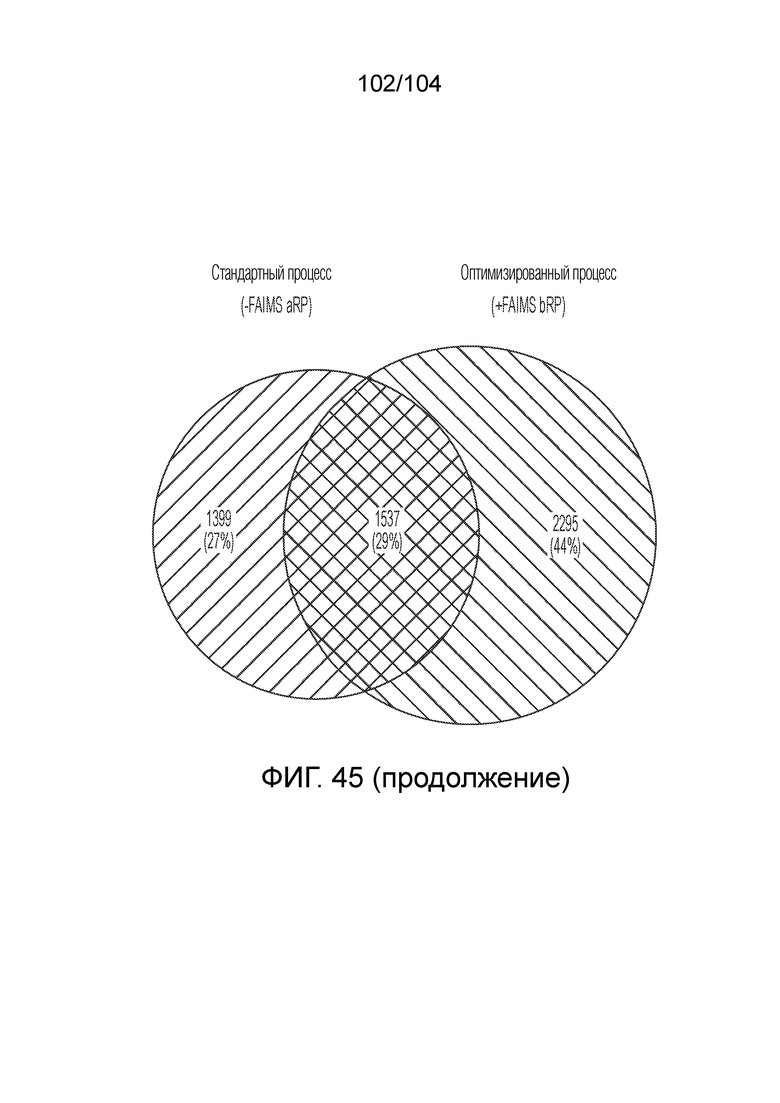
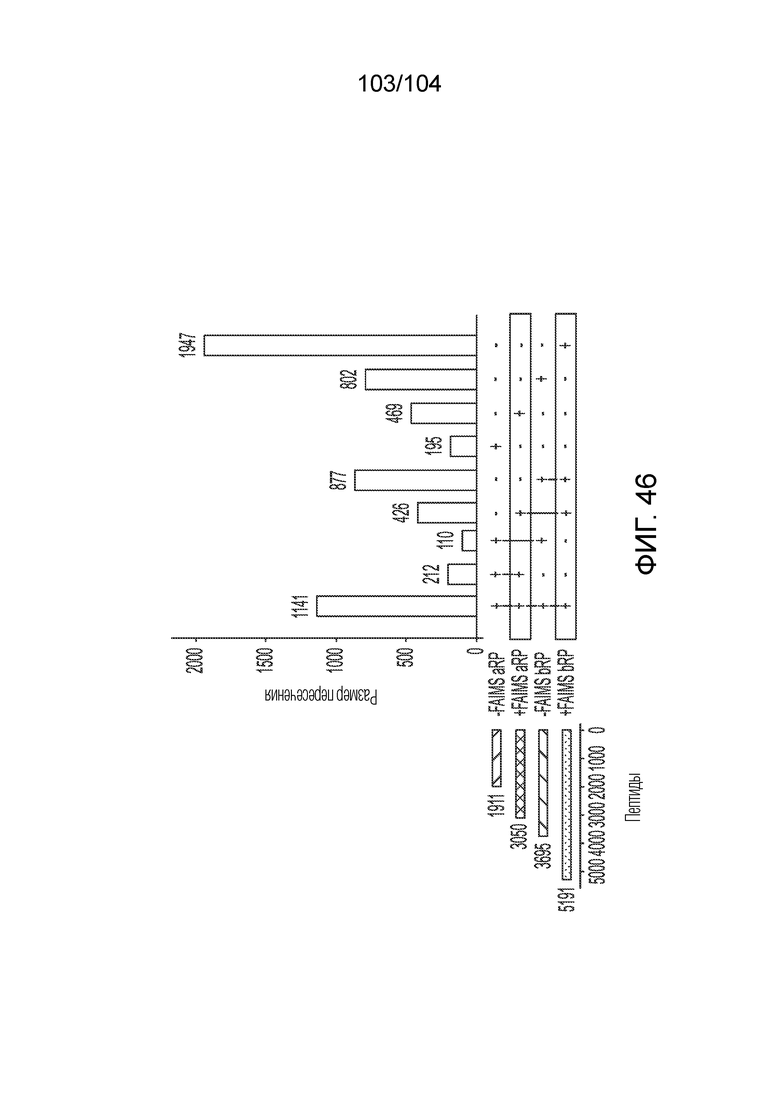
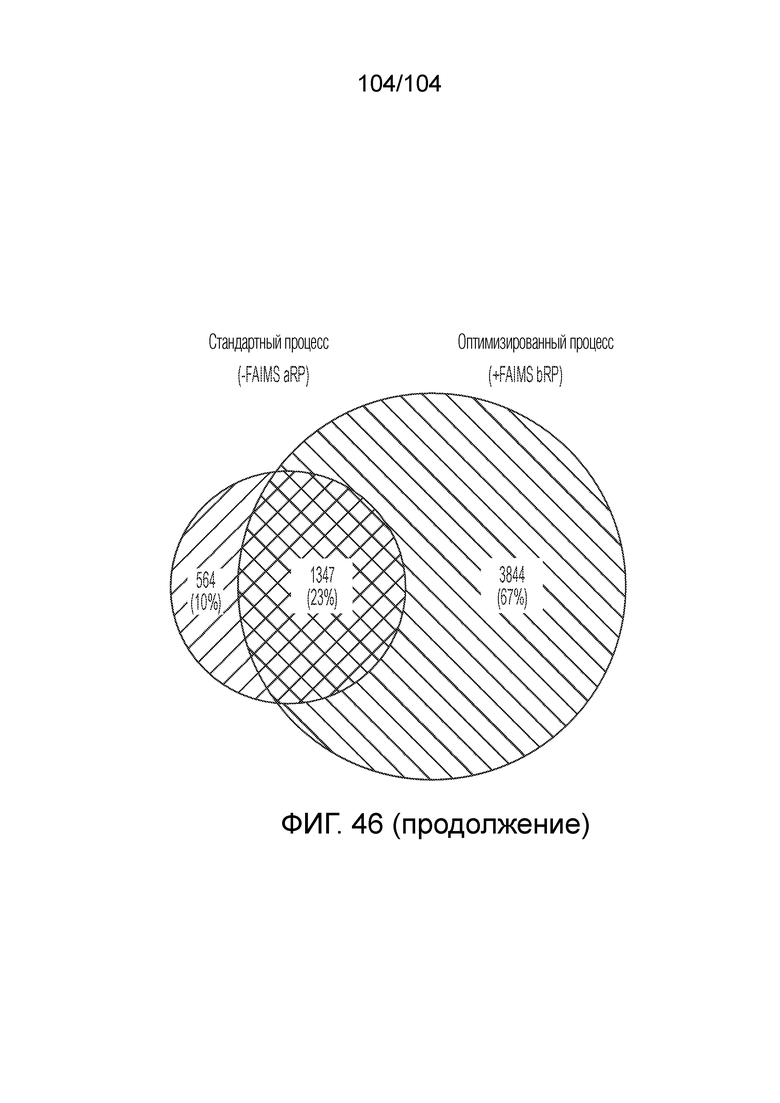
Изобретение относится к области биотехнологии. Описан способ приготовления персонализированной вакцины против злокачественной опухоли. Способ включает ввод информации об аминокислотах для набора последовательностей пептидов-кандидатов с использованием компьютерного процессора в обученную машинно-обучаемую модель для прогнозирования презентации пептидов HLA класса II с получением множества прогнозов презентации, выбор на основе по меньшей мере множества прогнозов презентации подгруппы пептидных последовательностей из набора последовательностей пептидов-кандидатов для получения набора выбранных пептидных последовательностей и приготовление персонализированной вакцины против злокачественной опухоли. Изобретение расширяет арсенал способов приготовления фармацевтической композиции с использованием обученной машинно-обучаемой модели. 18 з.п. ф-лы, 46 ил., 18 табл., 15 пр.
1. Способ приготовления персонализированной вакцины против злокачественной опухоли, включающий:
(a) ввод информации об аминокислотах для набора последовательностей пептидов-кандидатов с использованием компьютерного процессора в обученную машинно-обучаемую модель для прогнозирования презентации пептидов HLA класса II с получением множества прогнозов презентации, где каждая последовательность пептида-кандидата в наборе последовательностей пептидов-кандидатов кодируется геномом, транскриптомом или экзомом индивидуума, или патогеном или вирусом индивидуума; где множество прогнозов презентации включает прогноз презентации посредством HLA для каждой последовательности пептида-кандидата из набора последовательностей пептидов-кандидатов, где каждый прогноз презентации из множества прогнозов презентации указывает на вероятность того, что последовательность пептида из набора последовательностей пептидов-кандидатов презентируется одним или более белками, кодируемыми аллелем HLA класса II клетки индивидуума, и где обученная машинно-обучаемая модель для прогнозирования презентации пептидов посредством HLA класса II включает:
(i) множество параметров, идентифицированных по меньшей мере на основе обучающих данных, включающих:
(1) последовательности обучающих пептидов,
(2) идентичность белка, кодируемого аллелем HLA класса II, ассоциированным с обучающими пептидными последовательностями, и
(3) наблюдение посредством масс-спектрометрии презентации одного или более обучающих пептидов белком, кодируемым аллелем HLA класса II, в клетках для обучения; и
(ii) функцию, отражающую взаимосвязь между информацией об аминокислотной последовательности, полученной на входе, и вероятностью презентации в качестве результата, на основе информации об аминокислотной последовательности и множества параметров; и
(b) выбор на основе по меньшей мере множества прогнозов презентации подгруппы пептидных последовательностей из набора последовательностей пептидов-кандидатов для получения набора выбранных пептидных последовательностей, где выбранный набор пептидных последовательностей
(i) кодируется геномом, транскриптомом или экзомом пндивидуума, или патогеном или вирусом у индивидуума, и
(ii) спрогнозирован в качестве презентируемого белком, кодируемым аллелем HLA класса II клетки индивидуума;
(c) приготовление персонализированной вакцины против злокачественной опухоли, содержащей: (i) полипептид с одной или более из выбранной подгруппы пептидных последовательностей, (ii) полинуклеотид, кодирующий полипептид согласно (i), (iii) APC, содержащие (i) или (ii), или (iv) T-клетки, содержащие T-клеточный рецептор (TCR), специфический для белков, кодируемых аллелем HLA класса II индивидуума-человека, в комплексе с пептидной последовательностью, выбранной согласно (b), где обученная машинно-обучаемая модель для прогнозирования презентации пептидов посредством HLA имеет положительную прогностическую величину (PPV) по меньшей мере 0,2 при частоте отзыва 10% в соответствии со способом определения PPV презентации.
2. Способ по п.1, где каждая последовательность из последовательностей обучающих пептидов, выявленных посредством масс-спектрометрии в качестве презентируемых белком, кодируемым HLA класса II в клетках для обучения, имеет длину по меньшей мере 15 аминокислот.
3. Способ по п.1, где обученная машинно-обучаемая модель для прогнозирования презентации пептидов посредством HLA класса II имеет положительную прогностическую величину (PPV) по меньшей мере 0,3 в соответствии со способом определения PPV презентации.
4. Способ по п.3, где: (i) клетки для обучения включают клетки для обучения, экспрессирующие единичный комплекс MHC класса II или белок, кодируемый единичным аллельным вариантом локуса HLA класса II, выбранным из группы, состоящей из DR, DP и DQ, где единичный комплекс MHC класса II или белок, кодируемый единичным аллельным вариантом локуса HLA класса II, экспрессируется клеткой индивидуума; или (ii) обучающие данные включают обучающие данные, полученные посредством обратной свертки.
5. Способ по п.3, где клетки для обучения экспрессируют белок, кодируемый аллелем HLA класса II клетки индивидуума, где белок, кодируемый аллелем HLA класса II, содержит аффинную метку.
6. Способ по п.3, где машинно-обучаемая модель для прогнозирования презентации пептидов HLA имеет положительную прогностическую величину (PPV) по меньшей мере 0,2, когда информация об аминокислотах для множества тестовых пептидных последовательностей обрабатывается с получением множества тестовых прогнозов презентации, причем каждый тестовый прогноз презентации указывает на вероятность того, что один или более белков, кодируемых аллелем HLA класса II клетки индивидуума, может презентировать данную тестовую пептидную последовательность из множества тестовых пептидных последовательностей, где множество тестовых пептидных последовательностей включает по меньшей мере 500 тестовых пептидных последовательностей, включающих: (i) по меньшей мере одну наиболее подходящую пептидную последовательность, идентифицированную посредством масс-спектрометрии в качестве презентируемой белком HLA, экспрессируемым в клетках, и (ii) по меньшей мере 499 последовательностей пептидов-ловушек, содержащихся в белке, кодируемом геномом организма, где организм и индивидуум принадлежат одному виду, где множество тестовых пептидных последовательностей включает соотношение 1:499 для по меньшей мере одной наиболее подходящей последовательности и по меньшей мере 499 последовательностей пептидов-ловушек, и наивысшие 0,2% из множества тестовых пептидных последовательностей прогнозируются машинно-обучаемой моделью для прогнозирования презентации пептидов посредством HLA в качестве презентируемых белком HLA, экспрессируемым в клетках.
7. Способ по п.6, где указанное множество тестовых пептидных последовательностей содержит по меньшей мере 5000 тестовых пептидных последовательностей, содержащих по меньшей мере 10 наиболее подходящих пептидных последовательностей и по меньшей мере 4990 последовательностей пептидов-ловушек.
8. Способ по п.7, где любые подпоследовательности из девяти последовательно расположенных аминокислот любых из по меньшей мере 10 наиболее подходящих пептидных последовательностей не перекрываются с какими-либо подпоследовательностями из девяти последовательно расположенных аминокислот из по меньшей мере 4990 последовательностей пептидов-ловушек.
9. Способ по п.1, где каждая пептидная последовательность набора последовательностей пептидов-кандидатов ассоциирована со злокачественной опухолью.
10. Способ по п.9, где каждая пептидная последовательность из набора последовательностей пептидов-кандидатов (i) содержит мутацию, (ii) экспрессируется в злокачественной клетке индивидуума, и (iii) не кодируется геномом незлокачественной клетки индивидуума.
11. Способ по п.1, где белок, кодируемый аллелем HLA класса II, выбран из группы, состоящей из: HLA-DPB1*01:01/HLA-DPA1*01:03, HLA-DPB1*02:01/HLA-DPA1*01:03, HLA-DPB1*03:01/HLA-DPA1*01:03, HLA-DPB1*04:01/HLA-DPA1*01:03, HLA-DPB1*04:02/HLA-DPA1*01:03, HLA-DPB1*06:01/HLA-DPA1*01:03, HLA-DRB1*01:01, HLA-DRB1*01:02, HLA-DRB1*03:01, HLA-DRB1*03:02, HLA-DRB1*04:01, HLA-DRB1*04:02, HLA-DRB1*04:03, HLA-DRB1*04:04, HLA-DRB1*04:05, HLA-DRB1*04:07, HLA-DRB1*07:01, HLA-DRB1*08:01, HLA-DRB1*08:02, HLA-DRB1*08:03, HLA-DRB1*08:04, HLA-DRB1*09:01, HLA-DRB1*10:01, HLA-DRB1*11:01, HLA-DRB1*11:02, HLA-DRB1*11:04, HLA-DRB1*12:01, HLA-DRB1*12:02, HLA-DRB1*13:01, HLA-DRB1*13:02, HLA-DRB1*13:03, HLA-DRB1*14:01, HLA-DRB1*15:01, HLA-DRB1*15:02, HLA-DRB1*15:03, HLA-DRB1*16:01, HLA-DRB3*01:01, HLA-DRB3*02:02, HLA-DRB3*03:01, HLA-DRB4*01:01, HLA-DRB5*01:01, HLA-DRB1*01:01, HLA-DRB1*01:02, HLA-DRB1*03:01, HLA-DRB1*04:01, HLA-DRB1*04:02, HLA-DRB1*04:04, HLA-DRB1*04:05, HLA-DRB1*07:01, HLA-DRB1*08:01, HLA-DRB1*08:02, HLA-DRB1*08:03, HLA-DRB1*09:01, HLA-DRB1*11:01, HLA-DRB1*11:02, HLA-DRB1*11:04, HLA-DRB1*12:01, HLA-DRB1*13:01, HLA-DRB1*13:02, HLA-DRB1*13:03, HLA-DRB1*14:01, HLA-DRB1*15:01, HLA-DRB1*15:02, HLA-DRB1*15:03, HLA-DRB1*16:02, HLA-DRB3*01:01, HLA-DRB3*02:01, HLA-DRB3*02:02, HLA-DRB3*03:01, HLA-DRB4*01:01, HLA-DRB4*01:03, HLA-DRB5*01:01; HLA-DPB1*01:01, HLA-DPB1*02:01, HLA-DPB1*02:02, HLA-DPB1*03:01, HLA-DPB1*04:01, HLA-DPB1*04:02, HLA-DPB1*05:01, HLA-DPB1*06:01, HLA-DPB1*11:01, HLA-DPB1*13:01, HLA-DPB1*17:01, HLA-DQA1*01:01/HLA-DQB1*05:01, HLA-DQA1*01:02/HLA-DQB1*06:02, HLA-DQA1*01:02/HLA-DQB1*06:04, HLA-DQA1*01:03/HLA-DQB1*06:03, HLA-DQA1*02:01/HLA-DQB1*02:02, HLA-DQA1*02:01/HLA-DQB1*03:03, HLA-DQA1*03:01/HLA-DQB1*03:02, HLA-DQA1*03:03/HLA-DQB1*03:01, HLA-DQA1*05:01/HLA-DQB1*02:01, HLA-DQA1*05:05/HLA-DQB1*03:01 и их комбинации.
12. Способ по п.1, где способ включает получение множества полинуклеотидных последовательностей индивидуума посредством геномного, транскриптомного или экзомного секвенирования, где множество полинуклеотидных последовательностей кодируют набор последовательностей пептидов-кандидатов.
13. Способ по п.12, где геномное, транскриптомное или экзомное секвенирование представляет собой полногеномное секвенирование, полнотранскриптомное секвенирование или полноэкзомное секвенирование.
14. Способ по п.1, где набор последовательностей пептидов-кандидатов ранжированы на основе множества прогнозов презентации.
15. Способ по п.1, где каждая из пептидных последовательностей из набора выбранных пептидных последовательностей связывается с белком, кодируемым аллелем HLA класса II клетки индивидуума с IC50 500 нМ или менее или спрогнозированной IC50 500 нМ или менее.
16. Способ по п.1, где персонализированная вакцина против злокачественной опухоли содержит один или более полипептидов, содержащих по меньшей мере две из выбранных пептидных последовательностей, или один или более полинуклеотидов, кодирующих по меньшей мере две из выбранных пептидных последовательностей.
17. Способ по п.16, где персонализированная вакцина против злокачественной опухоли дополнительно содержит адъювант.
18. Способ по п.16, где персонализированная вакцина против злокачественной опухоли индуцирует ответ CD4+ T-клеток и/или ответ CD8+ T-клеток у индивидуума.
19. Способ по п.1, где обученная машинно-обучаемая модель для прогнозирования презентации пептидов посредством HLA класса II имеет положительную прогностическую величину (PPV) по меньшей мере 0,2 в соответствии со способом определения PPV презентации, при частоте отзыва 20%.
| US 20140178438 A1, 26.06.2014 | |||
| WO 2012159754 A2, 29.11.2012 | |||
| US 20170199961 A1, 13.07.2017 | |||
| WO 2018195357 A1, 25.10.2018 | |||
| Автоматические неравноплечие весы с передвижной при по мощи электродвигателя постоянного веса гирею | 1927 |
|
SU19358A1 |
| Polyakova A., Kuznetsova K., Moshkovskii S | |||
| Proteogenomics meets cancer immunology: mass spectrometric discovery and analysis of neoantigens //Expert review of proteomics, 2015, 12(5), p | |||
| ГИДРАВЛИЧЕСКИЙ ТАРАН | 1921 |
|
SU533A1 |
Авторы
Даты
2024-09-09—Публикация
2019-12-20—Подача