ПРИТЯЗАНИЕ НА ПРИОРИТЕТ
По данной заявке испрашивается приоритет на дату подачи временной патентной заявки США с серийным номером 61/390526, поданной 6 октября 2010 года, и безусловной патентной заявки с серийным номером 13/244049, поданной 23 сентября 2011 года, обе "MAIZE CYTOPLASMIC MALE STERILITY (CMS) C-TYPE RESTORER RF4 GENE, MOLECULAR MARKERS AND THEIR USE".
ТЕХНИЧЕСКАЯ ОБЛАСТЬ
Настоящее изобретение относится к генам фертильности растений. В некоторых вариантах осуществления изобретение относится к Rf4, гену-восстановителю фертильности кукурузы. В конкретных вариантах осуществления изобретение относится к композициям и способам восстановления фертильности при цитоплазматической мужской стерильности (CMS-C) C-типа, например, с использованием молекулярных маркеров, сцепленных с геном Rf4, или находящихся в нем. Конкретные варианты осуществления относятся к способам использования конкретных последовательностей нуклеиновых кислот для идентификации растений, которые содержат восстановитель фертильности CMS-C, и получения гибридных семян. Некоторые конкретные варианты осуществления относятся к полипептидам, ассоциированным с восстановлением фертильности CMS-C.
УРОВЕНЬ ТЕХНИКИ
Развитие разведения гибридных растений сделало возможными значительные достижения с точки зрения качества и количества продуцируемых культур. Увеличенный выход и комбинирование желаемых характеристик, таких как устойчивость к заболеваниям и насекомым, толерантность к жаре и засухе, и варьирование состава растения, являются возможными, частично, благодаря методикам гибридизации. Методики гибридизации основаны на вкладе, вносимом пыльцой из мужского родительского растения, в женское родительское растение, что приводит к получению гибрида.
Растения могут самоопыляться, если пыльца из одного цветка переносится на тот же или другой цветок одного и того же растения. Растения могут перекрестно опыляться, если пыльца происходит из цветка из другого растения. Растения кукурузы (Zea mays) можно выводить способами как самоопыления, так и перекрестного опыления. Растения кукурузы имеют мужские цветки, которые расположены на метелке, и женские цветки, которые расположены на початке того же растения. Естественное опыление кукурузы происходит, когда пыльца из метелки достигает рылец, которые находятся наверху зарождающихся початков. Разработка гибридов кукурузы основана на системах мужской стерильности.
Разработка гибридов кукурузы требует разработки гомозиготных инбредных линий, скрещивания этих линий и оценки продуктов скрещивания. Чистосортное разведение и рекуррентная селекция являются двумя способами разведения, используемыми для разработки инбредных линий из популяций. Программы разведения комбинируют желаемые признаки двух или более инбредных линий или различных универсальных источников в депо разведения, из которого разрабатывают новые инбредные линии путем самоопыления и селекции желаемых фенотипов. Гибридный сорт кукурузы является результатом скрещивания двух таких инбредных линий, каждая из которых может иметь одну или несколько желаемых характеристик, отсутствующих в одной, или дополняющих другую. Новые инбредные растения скрещивают с другими инбредными линиями, и гибриды этих скрещиваний оценивают для определения тех из них, которые являются желательными. Гибридное потомство первого поколения обозначают F1. При выведении гибридов искомыми являются только гибриды F1. Гибрид F1, как правило, является более сильным, чем его инбредные родительские растения. Эта сила гибридов, называемая гетерозисом, как правило, приводит, например, к повышенному вегетативному росту и увеличенному выходу.
Гибридную кукурузу можно получать с помощью системы мужской стерильности, включающей удаление метелки вручную. Для получения гибридных семян мужскую метелку удаляют из растущих женских инбредных родительских растений, которые можно высевать по принципу различным образом чередующихся рядов с мужскими инбредными родительскими растениями. Следовательно, при условии, что существует достаточная изоляция от чужеродной пыльцы, початки женского инбредного растения будут оплодотворяться только пыльцой мужского инбредного растения. Полученные семена представляют собой гибридные семена F1.
Удаление метелок вручную является трудоемким и дорогостоящим. Удаление метелок вручную часто является неэффективным, например, поскольку варьирование условий окружающей среды при выведении растений может приводить к растениям, у которых происходит выметывание метелок в женском родительском растении после проведения удаления метелок вручную, или поскольку лицо, осуществляющее удаление метелок, не полностью удалило метелку из женского инбредного растения. Если удаление метелок является неэффективным, женское растение будет продуктивно сбрасывать пыльцу, и некоторые женские растения будут самоопыляться. Это приведет к тому, что вместе с гибридными, семенами, продуцированными нормально, будут собраны семена женского инбредного растения. Семена женского инбредного растения являются не настолько продуктивными, как семена F1. Кроме того, присутствие женских инбредных семян может привести к риску недостаточной сохранности зародышевой плазмы для производителя гибридных семян.
Метелки из женского инбредного растения также можно удалять с помощью устройства. Механическое удаление метелок является приблизительно настолько же надежным, как и удаление метелок вручную, однако является более быстрым и менее дорогостоящим. Однако большинство устройств для удаления метелок приводит к большему повреждению растений, чем удаление метелок вручную. Таким образом, в настоящее время не существует полностью удовлетворительной формы удаления метелок.
Генетическая мужская стерильность является альтернативным способом, который можно использовать при получении гибридных семян. Трудоемкого процесса удаления метелок можно избежать в некоторых генотипах с использованием инбредных растений с цитоплазматической мужской стерильностью (CMS). В отсутствие гена-восстановителя фертильности, инбредные растения CMS обладают мужской стерильностью в результате факторов, происходящих из цитоплазматического, а не ядерного генома. Таким образом, характеристикой мужской стерильности является то, что она наследуется исключительно через женское растение в растениях кукурузы, поскольку только женское растение предоставляет цитоплазму для оплодотворенных семян. Растения CMS оплодотворяют пыльцой другого инбредного растения, которое не обладает мужской стерильностью. Пыльца второго инбредного растения может обеспечивать или может не обеспечивать гены, которые приводят к наличию у гибридных растений мужской фертильности. Обычно семена нормальной кукурузы после удаления метелок и продуцированные с помощью CMS семена того же гибрида должны быть смешаны для обеспечения того, чтобы достаточная нагрузка пыльцой была доступна для оплодотворения, когда гибридные растения выращивают, и для обеспечения цитоплазматического разнообразия.
Недостатки CMS как системы продуцирования гибридных семян включают ассоциацию конкретных вариантов CMS с предрасположенностью к определенным заболеваниям культур. См., например, Beckett (1971) Crop Science 11:724-6. Эта проблема, в частности, препятствует применению варианта CMS-T в продуцировании гибридных семян кукурузы и обычно негативно отражается на применении CMS в кукурузе.
Цитоплазматическая мужская стерильность (CMS) представляет собой наследуемую по материнской линии неспособность продуцировать функциональную пыльцу. Было найдено более 40 источников CMS, и они были классифицированы на три основных группы по различным реакциям восстановления фертильности в кукурузе. Эти группы обозначают как CMS-T (Texas), CMS-S (USDA) и CMS-C (Charrua). Beckett (1971). В группе CMS-T для восстановления фертильности пыльцы требуется два доминантных гена: Rf1 и Rf2, которые расположены на хромосомах 3 и 9, соответственно. Duvick (1965) Adv. Genetics 13:1-56. S-цитоплазма восстанавливается с помощью одного гена, Rf3, который картирован на 2 хромосоме. Laughnan and Gabay (1978) "Nuclear and cytoplasmic mutations to fertility in S male-sterile maize", Maize Breeding and Genetics, pp. 427-446.
В предшествующих анализах было обнаружено, что, по сравнению с CMS-T и CMS-S, восстановление фертильности CMS-C является очень сложным. Duvick (1972), "Potential usefulness of new cytoplasmic male sterile and sterility system", Proceeding of the 27th annual corn and sorghum research conference, pp. 197-201, обнаружил, что полное восстановление фертильности CMS-C контролируется доминантным аллелем гена Rf4. Khey-Pour et al. (1981) также обнаружили, что этот ген является достаточным для восстановления CMS-C. Однако Josephson et al. (1978), "Genetics and inheritance of fertility restoration of male sterile cytoplasms in corn", Proceedings of the 33rd com and sorghum research conference 7:13, предположили, что полное восстановление фертильности CMS-C обуславливается взаимодополняющим действием доминантных аллелей двух генов: Rf4 и Rf5, которые с тех пор были картированы на хромосомах 8 и 5, соответственно. Sisco (1991) Crop Sci. 31: 1263-6. Между тем, Chen et al. (1979) Acta Agronom. Sin. 5(4):21-28, предположили, что два доминантных гена-восстановителя при CMS-C имеют дублирующие функции. Далее усложняя систему, Vidakovic (1988), Maydica 33:51-65, продемонстрировал существование трех доминантных и комплементарных генов для полного восстановления фертильности при CMS-C, добавив ген Rf6. Vidakovic et al., (1997a) Maize Genet. Coop. News Lett. 71:10; (1997b) Maydica 42:313-6, позднее сообщили, что эти взаимодополняющие гены, Rf4, Rf5 и Rf6, в действительности не были единственными генетическими механизмами для восстановления фертильности при CMS-C в кукурузе. Таким образом, механизмы восстановления фертильности при CMS-C остаются не проясненными. В результате трудно выбрать линии-восстановители для некоторых генотипически стерильных линий.
Для ускорения процесса введения гена или локусов количественных признаков (QTL) в элитный культивар или линию выведения путем обратного скрещивания особенно пригодны молекулярные маркеры. Маркеры, сцепленные с геном, можно использовать для селекции растений, обладающих желаемым признаком, и маркеры по всему геному можно использовать для селекции растений, генетически сходных с рекуррентным родительским растением (Young and Tanksley (1989) Theor. Appl. Genet. 77:95-101; Hospital et al. (1992) Genetics 132:1199-210).
Большинство генов-восстановителей фертильности растений было клонировано с помощью стратегии клонирования на основе картирования. На сегодняшний день, было выделено девять генов Rf из нескольких видов растений, включая кукурузу (Zea Mays L.) (Cui et al. (1996) Science 272:1334-6; Liu et al. (2001) Plant Cell 13:1063-78), петунию (Petunia hybrida) (Bentolila et al. (2002) Proc. Natl. Acad. Sci. USA 99:10887-92, хрен (Raphanus sativus L.) (Brown et al. (2003) Plant J. 35:262-72; Desloire et al. (2003) EMBO Rep. 4:1-7; Koizuka et al. (2003) Plant J. 34:407-15), сорго (Sorghum bicolor L.) (Klein et al. (2005) Theor. Appl. Genet. 111: 994-1012), рис (Oryza sativa L.) (Kazama и Toriyama (2003) FEBS Lett. 544:99-102; Akagi et al. (2004) Theor. Appl. Genet. 108: 1449-57; Komori et al. (2004) Plant J. 37:315-25; Wang et al. (2006) Plant Cell 18:676-87; и Fujii and Toriyama (2009) Proc. Natl. Acad. Sci. USA 106(23): 9513-8), и губастик (Mimulus guttatus) (Barr and Fishman (2010) Genetics 184:455-65).
Все из идентифицированных генов-восстановителей, за исключением Rf2 в кукурузе и Rf17 в рисе, кодируют различные белки с пентатрикопептидными повторами (PPR). Геномы растений кодируют несколько сотен белков PPR, многие из которых вовлечены в регуляцию экспрессии генов органелл. Lurin et al. (2004) Plant Cell 16:2089-103; и Schmitz-Linneweber and Small (2008) Trends Plant Sci. 12:663-70. Белок PPR содержит от 2 до 27 повторов по 35 аминокислот, называемых мотивами PPR. Small and Peeters, (2000) Trends Biochem. Sci. 25(2):46-7. Предсказано, что белки PPR связываются с РНК (Delannoy et al. (2007) Biochemical Society Transactions 35:1643-7), и многие белки PPR нацеливаются на митохондрии, где расположены ассоциированные с CMS гены и продукты. Lurin et al. (2004), выше. Данные указывают на то, что белки PPR связываются непосредственно с транскриптами CMS. Akagi et al. (2004), выше; Gillman et al. (2007) Plant J. 49:217-27; и Kazama et al. (2008) Plant J. 55:619-28. Белки Rf снижают экспрессию ассоциированных с CMS транскриптов путем изменения их характера процессинга (Kazama & Toriyama (2003), выше), снижения стабильности РНК (Wang et al. (2006), выше; и Ohta et al. (2010) Plant Cell Rep. 29:359-69), или предотвращения их трансляции (Kazama et al. (2008), выше).
Дополнительная информация, касающаяся генов-восстановителей фертильности из кукурузы, риса, петунии и хрена, может быть найдена в патентной заявке США с серийным номером № US2006/0253931, и в патентах США № 5981833; 5624842; 4569152; 6951970; 6392127; 7612251; 7314971; 7017375; 7164058 и 5644066.
ОПИСАНИЕ ИЗОБРЕТЕНИЯ
В настоящем описании описано картирование локуса Rf4 кукурузы до небольшой области размером 12 т.п.н., расположенной в верхней части хромосомы 8. В этой области единственным вероятным кандидатом для Rf4 является ген, кодирующий фактор транскрипции bHLH. Путем клонирования локуса Rf4-bHLH из линий CMS-C, линий-невосстановителей и линий-восстановителей, был идентифицирован ряд различий в последовательности. На белковом уровне все из линий CMS-C и линий-невосстановителей имеют одну и ту же последовательность, и отличаются от аллелей-восстановителей (также идентичных друг другу) на 4 аминокислотных замены, включая консервативный гидрофильный остаток тирозина в домене bHLH (Y186), который заменен на гидрофобный остаток фенилаланина (F187) в линии-восстановителе.
Ген Rf4 кукурузы и кодируемый им полипептид представлены в настоящем описании, и кроме того, описаны молекулы нуклеиновых кислот, содержащие последовательность гена Rf4. Неожиданно, ген Rf4 не является геном белка с пентатрикопептидным повтором (PPR), как практически все другие гены-восстановители фертильности. Более того, показано, что восстановление фертильности в зародышевой плазме системы CMS-C/Rf4 по настоящему изобретению контролируется Rf4 в качестве единственного доминантного гена-восстановителя, что было неожиданным вследствие недавних работ нескольких групп. См. выше. Гидрофильный остаток тирозина в домене bHLH rf4-bHLH (Y186) кукурузы, который заменен на гидрофобный остаток фенилаланина (F187) в линиях восстановителей, является консервативным среди однодольных растений. Таким образом, идентификация гена Rf4 и маркеров гена Rf4 значительно способствовала разработке и широкому внедрению признака восстановления фертильности CMS-C в зародышевой плазме растений.
В вариантах осуществления мутация консервативного остатка тирозина в положении 186 rf4-bHLH на гидрофобный аминокислотный остаток (например, фенилаланин) ответственна за фенотип восстановителя в полипептиде Rf4-bHLH. Таким образом, в настоящем описании описаны гены Rf4-bHLH кукурузы или ортологи генов Rf4-bHLH кукурузы, которые кодируют гидрофобный аминокислотный остаток в этом положении (как определяют выравниванием последовательностей), где эти гены обеспечивают фенотип восстановителя CMS-C при включении в растение.
В настоящем описании описаны молекулярные маркеры нуклеиновых кислот, которые сцеплены (например, сцеплены; прочно сцеплены; или чрезвычайно прочно сцеплены) с геном Rf4 кукурузы или которые расположены в нем. В некоторых вариантах осуществления маркеры, которые сцеплены (например, сцеплены; прочно сцеплены; или чрезвычайно прочно сцеплены) с геном Rf4 кукурузы или которые расположены в нем, или саму последовательность гена Rf4 кукурузы, можно использовать для введения гена Rf4 кукурузы в организмы, например, растения (например, кукуруза и другие однодольные растения).
Также в настоящем описании описаны способы использования маркеров нуклеиновых кислот, которые сцеплены с геном Rf4 или расположены в нем, например и не ограничиваясь этим, для идентификации растений с функциональным геном-восстановителем для CMS C-типа; для введения Rf4 в новые генотипы растений (например, путем скрещивания с использованием маркера или генетической трансформации); и для получения гибридных семян от скрещиваний мужских растений, содержащих молекулярные маркеры нуклеиновых кислот, которые сцеплены с геном Rf4 или расположены в нем, и женских растений, несущих CMS C-типа.
Кроме того, описаны средства для восстановления фертильности кукурузы CMS-C, и средства для идентификации растений, имеющих ген восстановления фертильности кукурузы CMS-C. В некоторых примерах средства для восстановления фертильности кукурузы CMS-C могут представлять собой маркер, который сцеплен (например, сцеплен; прочно сцеплен; или чрезвычайно прочно сцеплен) с геном Rf4 кукурузы или который расположен в нем. В некоторых примерах средства для идентификации растений, имеющих ген восстановления фертильности кукурузы CMS-C, могут представлять собой зонд, который специфично гибридизуется с маркером, который сцеплен (например, сцеплен; прочно сцеплен; или чрезвычайно прочно сцеплен) с геном Rf4 или который расположен в нем.
Также в настоящем описании описаны способы, посредством которых можно получать гибридные семена путем скрещиваний мужского растения, содержащего молекулярные маркеры нуклеиновых кислот, которые сцеплены (например, сцеплены; прочно сцеплены; или чрезвычайно прочно сцеплены) или с геном Rf4 кукурузы или которые расположены в нем, и женского растения, имеющего CMS C-типа. Получение таких гибридных семян может приводить к экономии затрат вследствие ручного или механического устранения удаления метелок, и, кроме того, могут увеличить выход семян.
Кроме того, описаны способы использования молекул нуклеиновых кислот, описанных в настоящем описании, для идентификации гомологичных последовательностей Rf4 из видов растений, отличных от кукурузы (например, путем сравнения последовательностей). В некоторых вариантах осуществления систему CMS-C/Rf4 для получения гибридных семян создают способами инженерии в видах растений, отличных от кукурузы.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
На ФИГ.1 представлено 197 маркеров SNP, определенных как находящиеся в области гена Rf4, и их положения на физической карте.
На ФИГ.2 представлены тридцать четыре случайным образом отобранных рекомбинантных растения с их фенотипическими данными и соответствующими генетическими данными по 27 SNP-маркерам.
На ФИГ.3 представлены относительные положения маркеров SNP для гена Rf4 и генов в пределах области размером 1,5 млн.п.н. на 8 хромосоме.
На ФИГ.4 представлены относительные положения маркеров SNP для гена Rf4 и генов в пределах области размером 0,56 млн.п.н. и в пределах области размером 100 т.п.н. на 8 хромосоме.
На ФИГ.5 представлено точное картирование Rf4 в области размером 12 т.п.н. Буквами указаны генотипы: A=гомозиготный по BE4207 (CMS); H=гетерозиготный. Стрелками указаны левый и правый пограничные маркеры Rf4, и два наиболее важных рекомбинантных растения.
На ФИГ.6 представлено схематическое изображение геномной структуры аллеля Rf4-bHLH, демонстрирующее полную кодирующую область (от кодона инициации до стоп-кодона - 1,38 т.п.н.), 5'-UTR/промотор размером 1,1 т.п.н., и 3'-UTR/терминатор размером 0,75 т.п.н.
На ФИГ.7 представлено выравнивание последовательностей Rf4-bHLH из следующих генотипов кукурузы: B73; BE4207; B104; XJH58; BE9515; и MLW03. Положения кодона инициации трансляции, стоп-кодона и маркеров с DAS-CMS21 по DAS-CMS34, расположенных в гене, обозначены. Положения SNP и InDel затемнены.
На ФИГ.8 представлено выравнивание предсказанной последовательности кДНК Rf4-bHLH из следующих генотипов кукурузы: B73; BE4207; B104; XJH58; BE9515; и MLW03. Положения кодона инициации трансляции, стоп-кодона и маркеров для DAS-CMS22-25, 28-29 и 31, расположенных в кДНК, обозначены.
На ФИГ.9 представлено выравнивание предсказанных белковых последовательностей Rf4-bHLH. Положения консервативного домена bHLH, сигналов ядерной локализации (NLS) и положения соответствующих маркеров для DAS-CMS22, 23 и 28 обозначены. Замена Tyr на Phe в домене bHLH вызвана динуклеотидной заменой AC на TT в положении 747 (предсказанной последовательности кДНК B73), практически рядом с маркером DAS-CMS24 (см. ФИГ.8 и полиморфизм ID 54 в таблице 3).
На ФИГ.10 представлены данные, демонстрирующие профили экспрессии Rf4-HLH. L1=5 - слабые листья, L2=7 - слабые листья, L3=9 - слабые листья, T = метелки с развивающимися пыльниками и пыльцой, P = сбрасываемая пыльца. A = гомозиготный по BE4207, H = гетерозиготный, B = гомозиготный по XJH58. Данные соответствуют средним значениям для трех растений каждого генотипа для сегрегирующих F3 и для 1 растения каждого из родительских растений. Планки погрешностей соответствуют стандартному отклонению.
На ФИГ.11 представлено выравнивание Rf4-bHLH кукурузы (из восстановителя XJH58 и невосстановителя BE4207) с их ортологами из других однодольных видов. Положение консервативного домена bHLH подчеркнуто. Четыре аминокислотных замены между Rf4-bHLH XJH58 и Rf4-bHLH BE4207 обозначены.
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
Последовательности нуклеиновых кислот, приведенные в прилагаемом списке последовательностей, представлены с использованием стандартных буквенных обозначений для нуклеотидных оснований, как определено в 37 C.F.R. § 1.822. Показана только одна цепь каждой последовательности нуклеиновой кислоты, однако следует понимать, что при любой отсылке на представленную цепь включается комплементарная цепь. Для простоты при описании гена или локуса ген может быть описан с помощью мутантной формы гена (например, Rf4, в противоположность rf4), даже несмотря на то, что истинная последовательность может представлять собой форму гена дикого типа в соответствующем положении в геноме. Тем не менее понятно, что оба аллеля имеют различные последовательности, и из контекста понятно, какой аллель подразумевается. В прилагаемом списке последовательностей:
В SEQ ID NO: 1-197 представлены иллюстративные нуклеотидные последовательности маркеров, которые сцеплены (например, сцеплены; прочно сцеплены; или чрезвычайно прочно сцеплены) с геном Rf4 кукурузы или которые расположены в нем.
В SEQ ID NO: 198-211 представлены нуклеотидные последовательности в области приблизительно 0,56 млн.п.н. в верхней части 8 хромосомы кукурузы, в которой аллель Rf4 был первоначально картирован. SEQ ID NO: 203 представляет собой аллель Rf4-bHLH.
SEQ ID NO: 212-216 представляют собой иллюстративные различия нуклеотидных последовательностей между линиями CMS (BE4207) и восстановителя (XJH58).
В SEQ ID NO: 217 представлена нуклеотидная последовательность интервала приблизительно 12 т.п.н. из сорта кукурузы B73, в котором был точно картирован аллель Rf4.
В SEQ ID NO: 218 представлена нуклеотидная последовательность аллеля rf4-bHLH из сортов кукурузы B73 и BE4207.
В SEQ ID NO: 219 представлена нуклеотидная последовательность аллеля bHLH из сорта кукурузы B104.
В SEQ ID NO: 220 представлена нуклеотидная последовательность аллеля Rf4-bHLH из сортов кукурузы XJH58, BE9515 и MLW03.
В SEQ ID NO: 221 представлена нуклеотидная последовательность предсказанной кДНК rf4-bHLH из сортов кукурузы B73 и B4207.
В SEQ ID NO: 222 представлена нуклеотидная последовательность предсказанной кДНК bHLH из сорта кукурузы B104.
В SEQ ID NO: 223 представлена нуклеотидная последовательность предсказанной кДНК Rf4-bHLH из сортов кукурузы XJH58, BE9515 и MLW03.
В SEQ ID NO: 224 представлена аминокислотная последовательность предсказанного полипептида rf4-bHLH кукурузы.
В SEQ ID NO: 225 представлена аминокислотная последовательность предсказанного полипептида Rf4-bHLH.
В SEQ ID NO: 226 представлена аминокислотная последовательность предсказанного полипептида rf4-bHLH Brachypodium distachyon.
В SEQ ID NO: 227 представлена аминокислотная последовательность предсказанного полипептида rf4-bHLH Sorghum bicolor.
В SEQ ID NO: 228 представлена аминокислотная последовательность предсказанного полипептида rf4-bHLH Oryza sativa.
В SEQ ID NO: 229 и 230 представлены сигналы ядерной локализации (NLS) в Rf4-bHLH.
СПОСОБ(Ы) ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
I. Обзор нескольких вариантов осуществления
В настоящем описании описаны конкретные варианты осуществления генов, влияющих на мужскую фертильность в растениях: Rf4 кукурузы и прочно сцепленные с ними генетические маркеры, которые могут быть пригодны в различных системах контроля мужской фертильности. Более того, полиморфизм, присущий описанным прочно сцепленным генетическим маркерам, позволяет растениеводу отслеживать конкретный аллель гена, Rf4 или rf4, в сегрегирующей популяции. Ген Rf4 был первоначально картирован на 8 хромосоме в трех популяциях, полученных скрещиванием четырех культиваров кукурузы: BE4207×BE9515; BE4207×LW03F; и BE4207×XJH58. Точное картирование и клонирование на основе картирования были продемонстрированы в качестве примера в популяции BE4207×XJH58, в конечном итоге локализовав ген Rf4 в пределах приблизительно 12 т.п.н.
Восстановление цитоплазматической мужской стерильности (CMS) является стандартной сельскохозяйственной практикой при получении гибридных семян в течение многих лет. Использование гена-восстановителя фертильности (Rf) при цитоплазматической мужской стерильности упрощает программы продуцирования и снижает общие затраты путем полного устранения ручного или машинного удаления метелок. Однако полное преимущество применений генетики восстановления фертильности для цитоплазматической мужской стерильности C-типа в кукурузе для получения гибридных семян не было осуществлено, поскольку предшествующие исследования генетики восстановления фертильности для цитоплазматической мужской стерильности C-типа в кукурузе привели к противоречивым результатам.
Ввиду практической важности цитоплазматической мужской стерильности и восстановления фертильности пыльцы при получении гибридных семян кукурузы и необходимости диверсификации источника цитоплазмы, описано точное картирование гена-восстановителя Rf4 кукурузы для CMS-C до очень небольшой области с использованием молекулярных маркеров с помощью способа генотипирования KASPar™ и идентификация гена Rf4 кукурузы с помощью клонирования на основе картирования. Было открыто, что Rf4 является единственным доминантным геном-восстановителем для CMS-C в трех инбредных растениях кукурузы: BE9515, MLW03 и XJH58.
Rf4 первоначально был картирован с использованием маркеров SSR и SNP до области приблизительно 5,0 млн.п.н., начиная с маркера SSR umc-1075 до верхней части короткого плеча хромосомы 8. Была создана популяция для подтверждения F2 BE4207×XJH58 с 500 особями, и она была оценена в отношении фертильности в полевых условиях. Был проведен скрининг всего 197 маркеров SNP в популяции для подтверждения и было идентифицировано 104 рекомбинанта в пределах области размером 5,0 млн.п.н. Путем сравнения фенотипических показателей и генотипических данных для информативных рекомбинантных линий, ген Rf4 кукурузы был положительно идентифицирован в пределах области приблизительно 0,56 млн.п.н. (14 генов), и, вероятно, в пределах 100 т.п.н. (6 генов).
Таким образом, использование вариантов осуществления способов, описанных в настоящем описании, продемонстрировало, что ген Rf4 выбран из группы, состоящей из GRMZM2G 122853 (SEQ ID NO: 198); AC187051.4JFG005 (SEQ ID NO: 199); GRMZM2G122851 (SEQ ID NO: 200); GRMZM2G122850 (SEQ ID NO: 201); GRMZM2G582028 (SEQ ID NO: 202); GRMZM2G021276 (SEQ ID NO: 203); GRMZM2G381376 (SEQ ID NO: 204); GRMZM2G081127 (SEQ ID NO: 205); GRMZM2G085111 (SEQ ID NO: 206); GRMZM2G085038 (SEQ ID NO: 207); GRMZM2G317468 (SEQ ID NO: 208); GRMZM2G328030 (SEQ ID NO: 209); GRMZM2G029450 (SEQ ID NO: 210); и GRMZM2G077212 (SEQ ID NO: 211).
С использованием большой популяции для точного картирования размером приблизительно 5000 особей, локус Rf4 был картирован до небольшой области размером приблизительно 12 т.п.н., расположенной в верхней части 8 хромосомы. Тем самым, было продемонстрировано, что ген Rf4 выбран из группы, состоящей из перемещающегося генетического элемента растений [GRMZM2G582028 (SEQ ID NO: 202)] и фактора транскрипции основная-спираль-петля-спираль (bHLH) (GRMZM2G021276 (SEQ ID NO: 203)). Среди этих двух генов единственным вероятным кандидатом для Rf4 является фактор транскрипции основная спираль-петля-спираль (bHLH), геном Rf4 является GRMZM2G021276 (SEQ ID NO: 203). Таким образом, в конкретных вариантах осуществления ген Rf4 представляет собой GRMZM2G021276 (SEQ ID NO: 203), который иногда называют в настоящем описании как Rf4-bHLH. Понятно, что ген Rf4 также может представлять собой последовательность ДНК, которая кодирует тот же полипептид, что и ген Rf4-bHLH кукурузы, например, кодирующую последовательность SEQ ID NO: 203.
Локус bHLH был клонирован из линии CMS BE4207 кукурузы; линии B104 кукурузы; и трех линий восстановителей кукурузы: XJH58, BE9515 и MLW03. Был идентифицирован ряд изменений последовательностей между различными инбредными растениями. Следует отметить, что три линии-восстановителя имеют идентичные последовательности ДНК Rf4-bHLH, в то время как B73 и BE4207 (которые не содержат функциональный восстановитель Rf4) являются идентичными. Последовательность B104 является более сходной с аллелем BE4207/B73, чем с аллелем-восстановителем. На белковом уровне все из BE4207, B73 и B104 имеют одинаковую последовательность и отличаются от генного продукта аллеля-восстановителя на 4 аминокислотных замены, включая замену гидрофобным фенилаланином консервативного гидрофильного тирозина в домене bHLH.
В соответствии с функцией Rf4 в восстановлении фертильности пыльцы, аллель-восстановитель Rf4-bHLH экспрессируется специфически в развивающихся метелках (с пыльниками и пыльцой) растений, которые восстанавливают CMS-C. В линии CMS кукурузы BE4207 не происходит ни появление пыльников, ни развитие функциональной пыльцы. В результате в листьях и мужских репродуктивных тканях из растений BE4207 была выявлена очень низкая экспрессия rf4-bHLH или отсутствие его экспрессии. Поскольку B73 (инбредное растение, которое ни содержит цитоплазму CMS-C, ни восстанавливает CMS-C) имеет значительную экспрессию rf4-bHLH, маловероятно, что восстановление фертильности является следствием различий в уровне экспрессии между аллелем-восстановителем (Rf4-bHLH) и аллелем-невосстановителем (rf4-bHLH).
Полагают, что восстановление мужской фертильности является следствием различий в аминокислотной последовательности между продуктами генов аллеля-восстановителя и аллеля-невосстановителя. В частности, Y186 rf4-bHLH кукурузы расположен в первой спирали (Carretero-Paulet et al. (2010) Plant Physiol. 153: 1398-412; и Pires and Dolan (2010) Mol. Biol. Evol. 27:862-74) в связывающем домене ДНК bHLH, и этот остаток является абсолютно консервативным в B73 (не CMS, не восстановитель), BE4207 (CMS, не восстановитель) и в ортологах сорго, риса и Brachypodium. В трех линиях восстановителей кукурузы этот гидрофильный остаток заменен гидрофобным фенилаланином (F187). Такая неконсервативная замена может значительно изменить структуру спирали в домене bHLH и повлиять на связывание ДНК и последующую транскрипцию гена. Ввиду вышесказанного, авторы изобретения предсказали, что аллель rf4 из B104 не восстанавливает фертильность CMS-C, поскольку bHLH B104 имеет идентичную белковую последовательность с rf4-bHLH B73 и BE4207, включая консервативный тирозин в положении 186.
В некоторых вариантах осуществления молекулярные маркеры на основе Rf4-bHLH или прочно сцепленные высокопроизводительные молекулярные маркеры, описанные в настоящем описании, можно использовать для идентификации генотипов восстановителя Rf4, интрогрессии Rf4 в новые генотипы в кукурузе и других растениях для мужской конверсии, и удаления Rf4 из женских растений CMS. Имея маркеры и ген Rf4, в настоящее время возможно надежно перенести Rf4 в элитную зародышевую плазму и увеличить масштаб использования системы CMS-C/Rf4 для получения гибридных семян. Полная реализация этой системы может обеспечить значительные финансовые преимущества для сельскохозяйственной промышленности и потребителей ее продукции.
II. Термины
Обратное скрещивание: способы обратного скрещивания можно использовать для введения последовательности нуклеиновой кислоты в растения. Способ обратного скрещивания широко используют в течение десятилетий для введения новых признаков в растения. Jensen, N., Ed. Plant Breeding Methodology, John Wiley & Sons, Inc., 1988. В типичном протоколе обратного скрещивания исходный представляющий интерес сорт (рекуррентное родительское растение) скрещивают со вторым сортом (нерекуррентное родительское растение), который имеет представляющий интерес ген, подлежащий переносу. Затем полученное потомство этого скрещивания вновь скрещивают с рекуррентным родительским растением, и этот процесс повторяют до тех пор, пока не получают растение, где по существу все из желаемых морфологических и физиологических характеристик рекуррентного растения не появятся в преобразованном растении, в дополнение к перенесенному гену из нерекуррентного родительского растения.
Сцепленные, прочно сцепленные и чрезвычайно прочно сцепленные: как используют в рамках изобретения, сцепление между генами или маркерами относится к явлению, когда гены или маркеры на хромосоме демонстрируют поддающуюся измерению вероятность совместной передачи особям следующего поколения. Чем ближе два гена или маркера друг к другу, тем ближе к (1) эта вероятность. Таким образом, термин "сцепленный" может относиться к одному или нескольким генам или маркерам, которые передаются совместно с геном с вероятностью более 0,5 (которая ожидается, исходя из независимого распределения, где маркеры/гены расположены на различных хромосомах). Поскольку близкое расположение двух генов или маркеров на хромосоме прямо связано с вероятностью того, что гены или маркеры будут передаваться совместно особям в следующем поколении, термин "сцепленный" также может относиться в настоящем описании к одному или нескольким генам или маркерам, которые расположены в пределах приблизительно 2,0 млн.п.н. друг от друга на одной и той же хромосоме кукурузы. Таким образом, два "сцепленных" гена или маркера могут быть разделены приблизительно 2,1 млн.п.н.; 2,00 млн.п.н.; приблизительно 1,95 млн.п.н.; приблизительно 1,90 млн.п.н.; приблизительно 1,85 млн.п.н.; приблизительно 1,80 млн.п.н.; приблизительно 1,75 млн.п.н.; приблизительно 1,70 млн.п.н.; приблизительно 1,65 млн.п.н.; приблизительно 1,60 млн.п.н.; приблизительно 1,55 млн.п.н.; приблизительно 1,50 млн.п.н.; приблизительно 1,45 млн.п.н.; приблизительно 1,40 млн.п.н.; приблизительно 1,35 млн.п.н.; приблизительно 1,30 млн.п.н.; приблизительно 1,25 млн.п.н.; приблизительно 1,20 млн.п.н.; приблизительно 1,15 млн.п.н.; приблизительно 1,10 млн.п.н.; приблизительно 1,05 млн.п.н.; приблизительно 1,00 млн.п.н.; приблизительно 0,95 млн.п.н.; приблизительно 0,90 млн.п.н.; приблизительно 0,85 млн.п.н.; приблизительно 0,80 млн.п.н.; приблизительно 0,75 млн.п.н.; приблизительно 0,70 млн.п.н.; приблизительно 0,65 млн.п.н.; приблизительно 0,60 млн.п.н.; приблизительно 0,55 млн.п.н.; приблизительно 0,50 млн.п.н.; приблизительно 0,45 млн.п.н.; приблизительно 0,40 млн.п.н.; приблизительно 0,35 млн.п.н.; приблизительно 0,30 млн.п.н.; приблизительно 0,25 млн.п.н.; приблизительно 0,20 млн.п.н.; приблизительно 0,15 млн.п.н.; приблизительно 0,10 млн.п.н.; приблизительно 0,05 млн.п.н.; приблизительно 0,025 млн.п.н.; приблизительно 0,012 млн.п.н.; и приблизительно 0,01 млн.п.н. Конкретные примеры маркеров, которые "сцеплены" с Rf4, включают нуклеотидные последовательности в верхней части 8 хромосомы генома кукурузы, например, SEQ ID NO: 1-197; и маркеры, упоминаемые в настоящем описании как полиморфизмы ID №№ 1-106 (таблица 3).
Как используют в рамках изобретения, термин "прочно сцепленный" может относиться к одному или нескольким генам или маркерам, которые расположены в пределах приблизительно 0,5 млн.п.н. друг от друга на одной и той же хромосоме кукурузы. Таким образом, два "прочно сцепленных" гена или маркера могут быть разделены приблизительно 0,6 млн.п.н.; приблизительно 0,55 млн.п.н.; 0,5 млн.п.н.; приблизительно 0,45 млн.п.н.; приблизительно 0,4 млн.п.н.; приблизительно 0,35 млн.п.н.; приблизительно 0,3 млн.п.н.; приблизительно 0,25 млн.п.н.; приблизительно 0,2 млн.п.н.; приблизительно 0,15 млн.п.н.; приблизительно 0,12 млн.п.н.; приблизительно 0,1 млн.п.н.; и приблизительно 0,05 млн.п.н. Конкретные примеры маркеров, которые "прочно сцеплены" с Rf4, включают SEQ ID NO: 6-9; SEQ ID NO: 105; SEQ ID NO: 109; SEQ ID NO: 11; SEQ ID NO: 15; SEQ ID NO: 118-120; SEQ ID NO: 23; SEQ ID NO: 26; SEQ ID NO: 34; SEQ ID NO: 35; SEQ ID NO: 37; SEQ ID NO: 38; SEQ ID NO: 44; SEQ ID NO: 49; SEQ ID NO: 51; SEQ ID NO: 60; SEQ ID NO: 63; SEQ ID NO: 64; SEQ ID NO: 67; SEQ ID NO: 73; SEQ ID NO: 77; SEQ ID NO: 78; SEQ ID NO: 83; SEQ ID NO: 189-191; и SEQ ID NO: 97; и маркеры, упоминаемые в настоящем описании как полиморфизмы ID №№ 1-106 (таблица 3).
Как используют в рамках изобретения, термин "чрезвычайно прочно сцепленный" может относиться к одному или нескольким генам или маркерам, которые расположены в пределах приблизительно 100 т.п.н. друг от друга на одной и той же хромосоме кукурузы. Таким образом, два "чрезвычайно прочно связанных" гена или маркера могут быть разделены приблизительно 125 т.п.н.; приблизительно 120 т.п.н.; приблизительно 115 т.п.н.; приблизительно 110 т.п.н.; приблизительно 105 т.п.н.; 100 т.п.н.; приблизительно 95 т.п.н.; приблизительно 90 т.п.н.; приблизительно 85 т.п.н.; приблизительно 80 т.п.н.; приблизительно 75 т.п.н.; приблизительно 70 т.п.н.; приблизительно 65 т.п.н.; приблизительно 60 т.п.н.; приблизительно 55 т.п.н.; приблизительно 50 т.п.н.; приблизительно 45 т.п.н.; приблизительно 40 т.п.н.; приблизительно 35 т.п.н.; приблизительно 30 т.п.н.; приблизительно 25 т.п.н.; приблизительно 20 т.п.н.; приблизительно 15 т.п.н.; приблизительно 12 т.п.н.; приблизительно 10 т.п.н.; приблизительно 5 т.п.н.; и приблизительно 1 т.п.н. Конкретные примеры маркеров, которые "чрезвычайно прочно сцеплены" с Rf4, включают SEQ ID NO: 105; SEQ ID NO: 109; SEQ ID NO: 111; SEQ ID NO: 115; SEQ ID NO: 118-120; SEQ ID NO: 123; SEQ ID NO: 126; и SEQ ID NO: 134; и маркеры, упоминаемые в настоящем описании как полиморфизмы ID №№ 1-106 (таблица 3).
Сцепленные, прочно сцепленные и чрезвычайно прочно сцепленные генетические маркеры Rf4 могут быть пригодными в осуществляемых с помощью маркеров программах выведения для идентификации восстановителей для типов генов цитоплазматической мужской стерильности C-типа, и для выведения этого признака в сортах кукурузы.
Локус: как используют в рамках изобретения, термин "локус" относится к положению на геноме, который соответствует поддающейся измерению характеристике (например, признаку). Локус SNP определяют с помощью зонда, который гибридизуется с ДНК, находящейся в локусе.
Маркер: как используют в рамках изобретения, маркер относится к гену или нуклеотидной последовательности, которые можно использовать для идентификации растений, имеющих конкретный аллель, например, Rf4. Маркер может быть описан как изменение в данном геномном локусе. Генетический маркер может представлять собой короткую последовательность ДНК, такую как последовательность, окружающая изменение одной пары оснований (однонуклеотидный полиморфизм или "SNP"), или длинную последовательность, например, минисателлитный/простой повтор последовательности ("SSR"). "Маркерный аллель" относится к варианту маркера, который присутствует в конкретном растении.
Термин “маркер”, как используют в рамках изобретения, может относиться к клонированному сегменту хромосомной ДНК кукурузы (например, как определяют с помощью одной из SEQ ID NO: 1-197 или полиморфизмов ID №№ 1-106 (таблица 3)), и также может относиться или альтернативно относится к молекуле ДНК, которая комплементарна клонируемому сегменту хромосомной ДНК кукурузы (например, ДНК, комплементарная одной из SEQ ID NO: 1-197 или полиморфизмам ID №№ 1-106 (таблица 3)).
В некоторых вариантах осуществления присутствие маркера в растении можно выявлять с использованием зонда нуклеиновой кислоты. Зонд может представлять собой молекулу ДНК или молекулу РНК. РНК-зонды можно синтезировать с помощью средств, известных в данной области, например, с использованием молекулы ДНК-матрицы. Зонд может содержать всю или часть нуклеотидной последовательности маркера и дополнительную непрерывную нуклеотидную последовательность из генома кукурузы. Его называют в настоящем описании "непрерывным зондом". Дополнительную непрерывную нуклеотидную последовательность называют "вышележащей" или "нижележащей" относительно исходного маркера, в зависимости от того, расположена ли непрерывная нуклеотидная последовательность из хромосомы кукурузы на 5'- или 3'-стороне исходного маркера, как, в целом, понятно. Дополнительная непрерывная нуклеотидная последовательность может быть расположена между исходным маркером и областью размером 100 т.п.н. на 8 хромосоме генома кукурузы, которая расположена между положениями на карте 564922 и 601460. Таким образом, непрерывная нуклеотидная последовательность может быть расположена между исходным маркером и областью размером 12 т.п.н. на 8 хромосоме генома кукурузы, которая расположена между положениями на карте 86247 и 98188. Как общепризнано специалистами в данной области, способ получения дополнительной непрерывной нуклеотидной последовательности для включения в маркер, можно повторять практически без ограничений (он ограничен только длиной хромосомы), тем самым идентифицируя дополнительные маркеры вдоль хромосомы кукурузы. Все из описанных выше маркеров можно использовать в некоторых вариантах осуществления настоящего изобретения.
Последовательность олигонуклеотидного зонда можно получать синтетически или путем клонирования. Подходящие клонирующие векторы хорошо известны специалистам в данной области. Олигонуклеотид может быть меченым или немеченым. Существует широкое множество способов мечения молекул нуклеиновых кислот, включая, например и не ограничиваясь ими: мечение радиоактивной меткой путем ник-трансляции; использование случайных праймеров; удлинение концов с помощью концевой дезокситрансферазы; или сходные с ними, где используемые нуклеотиды являются меченными, например, радиоактивным 32P. Другие метки, которые можно использовать, включают, например и не ограничиваясь ими: флуорофоры; ферменты; субстраты ферментов; кофакторы ферментов; ингибиторы ферментов; и т.п. Альтернативно использование метки, которая обеспечивает поддающийся детекции сигнал, сама по себе или совместно с другими реакционноспособными веществами, можно заменять лигандами, с которыми связываются рецепторы, где рецепторы являются мечеными (например, описанными выше метками) для обеспечения поддающихся детекции сигналов, либо самостоятельно, либо совместно с другими реагентами. См., например, Leary et al. (1983) Proc. Natl. Acad. Sci. USA 80:4045-9.
Зонд может содержать нуклеотидную последовательность, которая не является непрерывной, для последовательности исходного маркера; этот зонд называют в настоящем описании "не являющимся непрерывным зондом". Последовательность не являющегося непрерывным зонда расположена достаточно близко к последовательности исходного маркера на геноме кукурузы, так что не являющийся непрерывным зонд является генетически сцепленным с тем же геном (например, Rf4). Например, в некоторых вариантах осуществления не являющийся непрерывным зонд может быть расположен в пределах 500 т.п.н.; 450 т.п.н.; 400 т.п.н.; 350 т.п.н.; 300 т.п.н.; 250 т.п.н.; 200 т.п.н.; 150 т.п.н.; 125 т.п.н.; 120 т.п.н.; 100 т.п.н.; 0,9 т.п.н.; 0,8 т.п.н.; 0,7 т.п.н.; 0,6 т.п.н.; 0,5 т.п.н.; 0,4 т.п.н.; 0,3 т.п.н.; 0,2 т.п.н.; или 0,1 т.п.н. от исходного маркера в геноме кукурузы.
Зонд может быть точной копией маркера, подлежащего выявлению. Зонд также может представлять собой молекулу нуклеиновой кислоты, содержащую, или состоящий из, нуклеотидную последовательность, которая по существу идентична клонированному сегменту хромосомной ДНК кукурузы (например, как определено в SEQ ID NO: 1-197 и полиморфизмах ID №№ 1-106 (таблица 3)). Как используют в рамках изобретения, термин "по существу идентичный" может относиться к нуклеотидным последовательностям, которые являются более чем на 85% идентичными. Например, по существу идентичная нуклеотидная последовательность может быть на 85,5%; 86%; 87%; 88%; 89%; 90%; 91%; 92%; 93%; 94%; 95%; 96%; 97%; 98%; 99%, или 99,5% идентична эталонной последовательности.
Зонд также может представлять собой молекулу нуклеиновой кислоты, которая является "специфично гибридизующейся" или "специфично комплементарной" точной копии маркера, подлежащего выявлению ("ДНК-мишень"). "Специфично гибридизующийся" и "специфично комплементарный" являются терминами, которые указывают на достаточную степень комплементарности, чтобы происходило стабильное и специфичное связывание между молекулой нуклеиновой кислоты и ДНК-мишенью. Молекула нуклеиновой кислоты не должна быть на 100% комплементарна ее последовательности-мишени, чтобы быть специфично гибридизующейся. Молекула нуклеиновой кислоты является специфично гибридизующейся, когда существует достаточная степень комплементарности, чтобы избежать неспецифического связывания нуклеиновой кислоты с не являющимися мишенями последовательностями в условиях, где специфичное связывание является желательным, например, в жестких условиях гибридизации.
Условия гибридизации, обеспечивающие конкретную степень жесткости, варьируют, в зависимости от выбранного способа гибридизации и композиции и длины гибридизующихся последовательностей нуклеиновых кислот. Как правило, температура гибридизации и ионная сила (особенно концентрация Na+ и/или Mg2+) буфера для гибридизации определяют жесткость гибридизации, хотя время промывания также влияет на жесткость. Вычисления, касающиеся условий гибридизации, требуемых для достижения конкретной степени жесткости, известны средним специалистам в данной области, и рассмотрены, например, в Sambrook et al. (ed.) Molecular Cloning: A Laboratory Manual, 2nd ed., vol. 1-3, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, 1989, chapters 9 и 11; и Hames and Higgins (eds.) Nucleic Acid Hybridization, IRL Press, Oxford, 1985. Дальнейшая детальная инструкция и руководство, касающиеся гибридизации нуклеиновых кислот, могут быть найдены, например, в Tijssen, "Overview of principles of hybridization and the strategy of nucleic acid probe assays", Laboratory Techniques in Biochemistry and Molecular Biology-Hybridization with Nucleic Acid Probes, Part I, Chapter 2, Elsevier, NY, 1993; и Ausubel et al., Eds., Current Protocols in Molecular Biology, Chapter 2, Greene Publishing and Wiley-Interscience, NY, 1995.
Как используют в рамках изобретения, "жесткие условия" охватывают условия, в которых гибридизация происходит, только если существует менее чем 25% несоответствие оснований между гибридизующейся молекулой ДНК-мишенью. "Условия жесткости" включают другие конкретные уровни жесткости. Таким образом, как используют в рамках изобретения, условия "умеренной жесткости" представляют собой условия, в которых молекулы с более чем 25% несоответствием последовательностей не будут гибридизоваться; условия "средней жесткости" представляют собой условия, в которых молекулы с более чем 15% несоответствием не будут гибридизоваться; и условия "высокой жесткости" представляют собой условия, в которых последовательности с более чем 10% несоответствием оснований не будут гибридизоваться. Условия "очень высокой жесткости" представляют собой условия, в которых последовательности с более чем 6% несоответствием не будут гибридизоваться.
В конкретных вариантах осуществления жесткие условия представляют собой гибридизацию при 65°C в 6× солевом натрий-цитратном буфере (SSC), 5× растворе Денхардта, 0,5% SDS и 100 г расщепленной ДНК семенников лосося, с последующими последовательными промываниями в течение 15-30 минут при 65°C в 2× буфере SSC и 0,5% SDS, а затем 1× буфере SSC и 0,5% SDS, и наконец, в 0,2× буфере SSC и 0,5% SDS.
Что касается всех рассмотренных выше зондов, зонд может содержать дополнительные последовательности нуклеиновых кислот, например, промоторы; сигналы транскрипции и/или последовательности векторов. Любой из рассмотренных выше зондов можно использовать для определения дополнительных маркеров, которые прочно сцеплены с геном, вовлеченным в восстановление фертильности кукурузы с цитоплазматической стерильностью C-типа (например, Rf4). Маркеры, идентифицированные таким образом, могут быть эквивалентными иллюстративным маркерам, указанным в настоящем описании и, таким образом, они находятся в объеме изобретения.
Выведение с помощью маркера: как используют в рамках изобретения, термин "выведение с помощью маркера" может относиться к подходу для выведения непосредственно одного или нескольких комплексных признаков (например, восстановитель фертильности CMS-C). В существующей практике, растениеводы предпринимают попытки к идентификации легко выявляемых признаков, таких как цвет цветка, вид оболочки семян, или варианты изоферментов, которые сцеплены с желаемым с сельскохозяйственной точки зрения признаком. Затем растениеводы наблюдают сельскохозяйственный признак в сегрегирующих выводимых популяциях путем наблюдения за сегрегацией легко выявляемого признака. Однако при выведении растений доступно очень мало из этих взаимосвязей сцепления.
Выведение с помощью маркеров обеспечивает экономичный по времени и затратам способ улучшения сортов растений. Несколько примеров применения выведения с помощью маркеров вовлекают использование маркеров изоферментов. См., например, Tanksley and Orton, eds. (1983) Isozymes in Plant Breeding and Genetics, Amsterdam: Elsevier. Одним примером является маркер изофермента, ассоциированный с геном устойчивости к нематодным паразитам в томате. Признак устойчивости, контролируемый геном, обозначаемым Mi, локализован на 6 хромосоме томата и очень прочно сцеплен с Aps1, изоферментом кислой фосфатазы. Использование маркера изофермента Aps1 для непрямой селекции по гену Mi обеспечило то преимущество, что сегрегацию в популяции стало возможно однозначно определять с помощью электрофоретических способов; маркер изофермента можно оценивать в ткани рассады, устраняя необходимость в поддержании растений до взрослого состояния; и кодоминантность аллелей маркера изофермента позволяет различение между гомозиготами и гетерозиготами. См. Rick (1983) в Tanksley and Orton, выше.
Функционально связанный: первая нуклеотидная последовательность функционально связана со второй последовательностью нуклеиновой кислоты, когда первая последовательность нуклеиновой кислоты находится в функциональной взаимосвязи со второй последовательностью нуклеиновой кислоты. Например, промотор функционально связан с кодирующей последовательностью, если промотор влияет на транскрипцию или экспрессию кодирующей последовательности. При рекомбинантной продукции, функционально связанные последовательности нуклеиновых кислот, как правило, являются непрерывными, и, когда это необходимо, объединяют две кодирующих белок области в одной рамке считывания (например, в полицистронной ORF). Однако нуклеиновые кислоты не должны быть соседними, чтобы быть функционально связанными.
Промотор: как используют в рамках изобретения, термин "промотор" относится к области ДНК, которая может лежать выше точки начала транскрипции, и которая может быть вовлечена в распознавание и связывание РНК-полимеразы и других белков для инициации транскрипции. Промотор может быть функционально связан с геном для экспрессии в клетке, или промотор может быть функционально связан с нуклеотидной последовательностью, кодирующей сигнальную последовательность, которая может быть функционально связана с геном, для экспрессии в клетке. "Промотор растений" может представлять собой промотор, способный инициировать транскрипцию в клетках растений. Примеры промоторов, находящихся контролем стадии развития, включают промоторы, которые предпочтительно инициируют транскрипцию в определенных тканях, таких как листья, корни, семена, волокна, сосудах ксилемы, трахеидах или склеренхиме. Такие промоторы обозначают как "предпочтительные для тканей". Промоторы, которые инициируют транскрипцию только в определенных тканях, называют "тканеспецифическими". "Специфичный к типу клеток" промотор, главным образом, запускает экспрессию в определенных типах клеток в одном или нескольких органах, например, сосудистых клетках в корнях или листьях. "Индуцируемый" промотор может представлять собой промотор, который может находиться под контролем условий окружающей среды. Примеры условий окружающей среды, которые могут инициировать транскрипцию с помощью индуцибельных промоторов, включают анаэробные условия или присутствие света. Тканеспецифические, предпочтительные для тканей, специфичные к типу клеток и индуцибельные промоторы составляют класс "неконститутивных" промоторов. "Конститутивный" промотор представляет собой промотор, который может быть активным в большинстве условий окружающей среды.
В некоторых вариантах осуществления настоящего изобретения можно использовать любой индуцибельный промотор. См. Ward et al. (1993) Plant Mol. Biol. 22:361-366. В случае индуцибельного промотора скорость транскрипции возрастает в ответ на индуцирующий агент. Иллюстративные индуцибельные промоторы включают, но не ограничиваются ими: промоторы системы ACEI, которые отвечают на медь; ген In2 из кукурузы, который отвечает на антидоты для бензолсульфонамидных гербицидов; репрессор Tet из Tn10; и индуцибельный промотор из гена стероидного гормона, транскрипционную активность которого может индуцировать глюкокортикостероидный гормон (Schena et al. (1991) Proc. Natl. Acad. Sci. USA 88:0421).
Иллюстративные конститутивные промоторы включают, но не ограничиваются ими: промоторы из вирусов растений, такие как промотор 35S из CaMV; промоторы из генов актина риса; промоторы убиквитина; pEMU; MAS; промотор гистона H3 кукурузы; и промотор ALS, фрагмент XbaI/NcoI, находящийся с 5'-стороны от структурного гена ALS3 Brassica napus (или нуклеотидная последовательность, сходная с указанным фрагментом XbaI/NcoI) (международная заявка PCT WO 96/30530).
Любой тканеспецифический или предпочтительный для ткани промотор также можно использовать в некоторых вариантах осуществления настоящего изобретения. Растения, трансформированные геном, функционально связанным с тканеспецифическим промотором, могут продуцировать белковый продукт трансгена исключительно, или предпочтительно, в конкретной ткани. Иллюстративные тканеспецифические или предпочтительные для тканей промоторы включают, но не ограничиваются ими: предпочтительный для корней промотор, такой как промотор из гена фазеолина; специфичный для листьев и индуцируемый светом промотор, такой как промотор из cab или rubisco; специфичный для пыльника промотор, такой как промотор из LAT52; специфичный для пыльцы промотор, такой как промотор из Zm13; и предпочтительный для микроспор промотор, такой как промотор из apg.
Идентичность последовательностей: термин "идентичность последовательностей" или "идентичность", как используют в рамках изобретения в контексте двух последовательностей нуклеиновых кислот или полипептидов, может относиться к остаткам в двух последовательностях, которые являются одинаковыми при выравнивании на максимальное соответствие на протяжении определенного окна сравнения.
Когда процент идентичности последовательностей используют в отношении белков, является общепризнанным, что остатки, которые не являются идентичными, часто отличаются посредством консервативных аминокислотных замен, где аминокислотные остатки замещают другие аминокислотные остатки со сходными химическими свойствами (например, заряд, гидрофобность или пространственные эффекты), и, таким образом, не изменяют функциональных свойств молекулы.
Таким образом, когда последовательности отличаются консервативными заменами, процентная идентичность последовательностей может быть скорректирована в направлении увеличения для поправки на консервативную природу замены в области неидентичного остатка. Последовательности, которые отличаются такими консервативными заменами, называют имеющими "сходство последовательностей" или "сходство". Способы осуществления этой коррекции хорошо известны средним специалистам в данной области. Как правило, такие способы вовлекают оценку консервативной замены как частичное, а не полное, несоответствие, тем самым увеличивая процентную идентичность последовательностей. Например, когда идентичной аминокислоте присваивается показатель от 0 до 1, и неконсервативной замене присваивается показатель 0, консервативной замене присваивается показатель от 0 до 1. Оценку консервативных замен можно проводить, например, как осуществлено в программе PC/GENE (Intelligenetics, Mountain View, CA).
Как используют в рамках изобретения, термин "процент идентичности последовательностей" может относиться к величине, определяемой путем сравнения двух оптимально выровненных последовательностей на протяжении окна сравнения, где часть последовательности в окне сравнения может содержать вставки или делеции (т.е. пропуски) по сравнению с эталонной последовательностью (которая не содержит вставки или делеции) для оптимального выравнивания двух последовательностей. Процент вычисляют путем определения количества положений, в которых идентичный нуклеотидный или аминокислотный остаток встречается в обеих последовательностях, с получением соответствующих друг другу положений, деления количества соответствующих друг другу положений на общее число положений в окне сравнения, и умножения результата на 100 с получением процента идентичности последовательностей.
Однонуклеотидный полиморфизм (SNP): как используют в рамках изобретения, термин "однонуклеотидный полиморфизм" может относиться к варьированию последовательности ДНК, возникающему, когда отдельный нуклеотид в геноме (или другой общей последовательности) отличается между представителями вида или парными хромосомами индивидуума.
В популяции SNP может быть приписана частота редкого аллеля - наиболее низкая аллельная частота в локусе, которую наблюдают в конкретной популяции. Она представляет собой просто наименьшую из двух аллельных частот однонуклеотидных полиморфизмов. Существует варьирование между популяциями человека, так что аллель SNP, который является распространенным в одной географической или этнической группе, может быть значительно более редким в другой.
Однонуклеотидные полиморфизмы могут находиться в кодирующих последовательностях генов, некодирующих областях генов, или в межгенных областях между генами. SNP в кодирующей последовательность необязательно изменяет аминокислотную последовательность белка, который продуцируется, вследствие вырожденности генетического кода. SNP, в котором обе формы приводят к одной и той же полипептидной последовательности, называют "синонимическими" (иногда называемыми молчащей мутацией). Если продуцируется отличающаяся полипептидная последовательность, ее называют "несинонимической". Несинонимическая замена может представлять собой либо миссенс-замену, либо нонсенс-замену, где миссенс-замена приводит к отличающейся аминокислоте, а нонсенс-замена приводит к преждевременному стоп-кодону. SNP, которые не являются кодирующими белок областями, тем не менее, могут иметь последствия для сплайсинга генов, связывания факторов транскрипции или последовательности некодирующей РНК. SNP обычно являются биаллельными и, таким образом, их легко анализировать в растениях и животных. Sachidanandam (2001) Nature 409:928-33.
InDel: как используют в рамках изобретения, термин "InDel" используют, главным образом, для описания инсерции или делеции в гене. Таким образом, "InDel" просто относится к конкретной мутации, которая может представлять собой либо инсерцию, либо делецию, либо их комбинацию.
Признак или фенотип: термины "признак" и "фенотип" используют в настоящем описании взаимозаменяемо. Для целей настоящего изобретения представляющим особый интерес признаком является восстановление фертильности CMS C-типа.
III. Ген-восстановитель CMS-C кукурузы Rf4 и его молекулярные маркеры
Предусматриваются молекулярные маркеры, которые сцеплены (например, прочно сцеплены) с геном-восстановителем CMS-C кукурузы Rf4. Идентифицированы сегменты ДНК, содержащие последовательности, вовлеченные в восстановление фертильности растений CMS-C. Эти сегменты расположены между маркерами, которые сцеплены (например, прочно сцеплены) с геном Rf4. Таким образом, также предусмотрены молекулы нуклеиновых кислот, содержащие ген Rf4. Идентифицированные сегменты и их маркеры описаны в настоящем описании, частично по их положению в конкретной области в верхней части хромосомы 8 кукурузы.
Положение идентифицированных сегментов и их маркеров, когда его выражают в качестве частот рекомбинации или единиц картирования, предусмотрено в настоящем описании в качестве общей информации. Варианты осуществления, описанные в настоящем описании, осуществляли в популяции кукурузы BE4207×XJH58. Однако положения конкретных сегментов и маркеров в качестве единиц маркеров выражены в отношении общедоступной геномной последовательности инбредного растения кукурузы B73 (B73 RefGen v1 или v2), которая может быть найдена во всемирной сети на www2.genome.arizona.edu/genomes/maize, или ftp.maizesequence.org/current/assembly/. Последовательности генов сортов кукурузы BE4207 и XJH58 еще не доступны. Ожидается, что числа, приведенные для конкретных сегментов и маркеров в качестве единиц картирования, могут варьировать от культивара к культивару и не являются частью основного определения сегментов и маркеров ДНК, и эти сегменты и маркеры ДНК иным образом описывают, например, по нуклеотидной последовательности.
Доминантный аллель гена Rf4 контролирует восстановление фертильности в системе CMS-C/Rf4. В вариантах осуществления ген Rf4 определяют как ген, выбранный из группы, состоящей из GRMZM2G122853 (SEQ ID NO: 198); AC187051.4_FG005 (SEQ ID NO: 199); GRMZM2G122851 (SEQ ID NO: 200); GRMZM2G122850 (SEQ ID NO: 201); GRMZM2G582028 (SEQ ID NO: 202); GRMZM2G021276 (SEQ ID NO: 203); GRMZM2G381376 (SEQ ID NO: 204); GRMZM2G081127 (SEQ ID NO: 205); GRMZM2G085111 (SEQ ID NO: 206); GRMZM2G085038 (SEQ ID NO: 207); GRMZM2G317468 (SEQ ID NO: 208); GRMZM2G328030 (SEQ ID NO: 209); GRMZM2G029450 (SEQ ID NO: 210); и GRMZM2G077212 (SEQ ID NO: 211). В конкретных вариантах осуществления ген Rf4 представляет собой Rf4-bHLH (SEQ ID NO: 203). Например, ген Rf4-bHLH представлен в SEQ ID NO: 220.
В некоторых вариантах осуществления изобретение также включает нуклеотидные последовательности, которые по существу идентичны Rf4-bHLH. Например, в некоторых вариантах осуществления молекула нуклеиновой кислоты представляет собой гомолог Rf4, который по меньшей мере приблизительно на 85% идентичен Rf4-bHLH. Гомолог Rf4 может быть на 86%; 87%; 88%; 89%; 90%; 91%; 92%; 93%; 94%; 95%; 96%; 97%; 98%; 99%, или 99,5% идентичен Rf4-bHLH. Такой гомолог Rf4 можно легко идентифицировать и выделять из полных или частичных генов, легкодоступных специалистам в данной области для множества организмов.
Некоторые варианты осуществления также включают функциональные варианты гена Rf4. Функциональные варианты Rf4 включают, например, последовательность Rf4-bHLH, содержащую одну или несколько нуклеотидных замен, делеций или вставок, где функциональный вариант восстанавливает мужскую фертильность в кукурузе CMS-C, как можно определять общепринятыми способами, хорошо известными специалистам в данной области. Например, способность конкретного варианта гена Rf4 восстанавливать мужскую фертильность кукурузы CMS-C можно определять путем общепринятого внесения мутации или фрагмента в растения, гомозиготные по стерильному аллелю rf4, с последующим стандартным наблюдением растений в отношении мужской стерильности. Функциональные варианты гена Rf4 можно создавать с помощью сайт-направленного мутагенеза, индуцируемой мутации или они могут встречаться в качестве аллельных вариантов (полиморфизмы, например, SNP). В конкретных примерах функциональный вариант Rf4 представляет собой последовательность Rf4-bHLH, содержащую одну или несколько нуклеотидных замен, делеций или вставок, так что вариант кодирует полипептид Rf4-bHLH, содержащий гидрофобную аминокислотную замену (например, Phe) для Y186 в домене bHLH.
Таким образом, в некоторых вариантах осуществления функциональные варианты гена Rf4 могут представлять собой мутации Rf4, или фрагменты, меньшие, чем полная последовательность Rf4, которые сохраняют свойства контроля мужской стерильности гена Rf4. Такие мутации и фрагменты, таким образом, считаются находящимися в объеме изобретения. Ввиду настоящего описания, специалист в данной области может легко определить, сохраняет ли мутация или фрагмент последовательности Rf4, указанной в настоящем описании, свойства гена Rf4.
В некоторых вариантах осуществления изобретение также включает полипептиды Rf4-bHLH (например, SEQ ID NO: 225) и полипептиды, которые по существу идентичны Rf4-bHLH. Например, в некоторых вариантах осуществления полипептид, который по существу идентичен Rf4-bHLH, может быть по меньшей мере приблизительно на 25% идентичен Rf4-bHLH и имеет гидрофобный аминокислотный остаток (например, Phe) в положении, соответствующем F187 SEQ ID NO: 225, при определении с помощью выравнивания последовательностей. В некоторых вариантах осуществления полипептид, который по существу идентичен Rf4-bHLH, может быть на 86%; 87%; 88%; 89%; 90%; 91%; 92%; 93%; 94%; 95%; 96%; 97%; 98%; 99%, или 99,5% идентичен Rf4-bHLH. Такие полипептиды, которые по существу идентичны Rf4-bHLH, можно легко идентифицировать и установить из полных или частичных геномов или библиотек кДНК, свободно доступных специалистам в данной области для множества организмов.
IV. Способы использования гена Rf4
Ген Rf4, описанный в настоящем описании, можно использовать в любом из множества способов, известных специалисту в данной области, для манипуляции геном, чтобы обеспечить желаемый эффект. Например и не ограничиваясь этим, ген Rf4 можно использовать для: внесения мутантной последовательности Rf4 в растение для обеспечения стерильности; внесения мутации в нативную последовательность Rf4; внесения антисмысловой молекулы нуклеиновой кислоты, нацеленной на ДНК или РНК Rf4, в растение для влияния на фертильность; использования образования шпилек; или связывания последовательности(ей) Rf4 с другими последовательностями нуклеиновой кислоты для контроля экспрессии продукта гена Rf4.
Например, в некоторых вариантах осуществления ген Rf4, определенный как выбранный из группы, состоящей из GRMZM2G122853 (SEQ ID NO: 198); AC187051.4_FG005 (SEQ ID NO: 199); GRMZM2G122851 (SEQ ID NO: 200); GRMZM2G122850 (SEQ ID NO: 201); GRMZM2G582028 (SEQ ID NO: 202); GRMZM2G021276 (SEQ ID NO: 203); GRMZM2G381376 (SEQ ID NO: 204); GRMZM2G081127 (SEQ ID NO: 205); GRZM2G085111 (SEQ ID NO: 206); GRMZM2G085038 (SEQ ID NO: 207); GRMZM2G317468 (SEQ ID NO: 208); GRMZM2G328030 (SEQ ID NO: 209); GRMZM2G029450 (SEQ ID NO: 210); и GRMZM2G077212 (SEQ ID NO: 211) можно использовать для облегчения использования системы мужской фертильности CMS-C/Rf4 совместно с другими генами или мутантами, влияющими на мужскую фертильность в кукурузе. Например, в конкретных вариантах осуществления ген Rf4-bHLH можно использовать для облегчения использования системы мужской фертильности CMS-C/Rf4 совместно с другими генами или мутантами, влияющими на мужскую фертильность в кукурузе.
В некоторых вариантах осуществления ген Rf4 можно вносить в растение кукурузы, которое является пригодным для применения в системе мужской фертильности, отличной от системы мужской фертильности CMS-C/Rf4. Альтернативно ген или мутантный ген, отличный от Rf4, можно вносить в растение кукурузы, которое пригодно для применения в системе мужской фертильности CMS-C/Rf4, так чтобы внесенный ген или мутантный ген можно было использовать для обеспечения дополнительного или дополняющего контроля фертильности. Конкретные примеры других генов мужской фертильности и мутаций в кукурузе включают: CMS-T/Rf1; CMS-T/Rf2; CMS-S/Rf2; ms1 (Singleton and Jones (1930) J. Hered. 21: 266-8); ms2 и ms3 (Eyster (1931) J. Hered. 22:99-102); ms5, ms7, ms8, ms9, ms10, ms11, ms12, ms13 и ms14 (Beadle (1932) Genetics 17:413-31); ms17 (Emerson (1932) Science 75:566); ms20 (Eyster (1934) Bibliographia Genetica 11:187-392); ms23 и ms24 (West and Albertsen (1985) MNL 59:87); ms25 и ms26 (Loukides et al. (1995) Am. J. Bot. 82:1017-23); ms27 и ms38 (Albertsen et al. (1996) MNL 70:30-1); ms28 (Golubovskaya (1979) MNL 53:66-70); ms29 и ms31 (Trimnell et al. (1998) MNL 72:37-38); ms30 (Albertsen et al. (1999) MNL 73:48); ms32, ms36 и ms37 (Trimnell et al. (1999) MNL 73:48-50); ms33 и ms34 (Patterson (1995) MNL 69:126-8); ms43 (Golubovskaya (1979) Int. Rev. Cytol. 58:247-90); ms45 (Albertsen et al. (1993) Proc. Annu. Corn Sorghum Ind. Res. Conf. 48:224-33) и ms48, ms49 и ms50 (Trimnell et al. (2002) MNL 76:38-9).
Когда последовательность нуклеиновой кислоты (например, Rf4) "вводят" в организм, такой как растение, способ или методология, используемые для введения молекулы нуклеиновой кислоты, содержащей конкретную последовательность, не являются существенными для изобретения, и его можно проводить с помощью любого способа или методологии, известных специалистам в данной области. Например, молекулу нуклеиновой кислоты можно вводить способами прямой трансформации, такими как опосредуемая Agrobacterium трансформация ткани растений; бомбардировка микроснарядами; электропорация; и т.д. Альтернативно молекулу нуклеиновой кислоты можно вводить путем скрещивания растения, имеющего конкретную нуклеотидную последовательность, с другим растением, так чтобы потомство имело нуклеотидную последовательность, включенную в их геном. Такие способы выведения хорошо известны специалисту в данной области. Способы выведения с помощью маркера, как описано в настоящем описании, могут значительно облегчать включение Rf4 путем таких скрещиваний.
В вариантах осуществления, где ген Rf4 вводят в организм, может быть желательным введение гена Rf4 таким образом, чтобы ген Rf4 был функционально связан с одной или несколькими регуляторными последовательностями, например, введение с использованием плазмиды, содержащей ген Rf4, функционально связанный с желаемыми регуляторными последовательностями. Регуляторные последовательности, пригодные для экспрессии гетерологичных последовательностей нуклеиновых кислот, хорошо известны в данной области, и включают, например и не ограничиваясь ими: промоторы (например, конститутивные промоторы; тканеспецифические промоторы; и специфические для стадии развития промоторы); последовательности терминации; энхансерные последовательности; последовательности субклеточного нацеливания; стабилизирующие или лидерные последовательности; и интроны.
В некоторых вариантах осуществления ген Rf4 можно вводить в организм с одной или несколькими дополнительными желаемыми последовательностями нуклеиновых кислот (например, генами). Дополнительные желаемые последовательности нуклеиновых кислот могут включать, например: гены, кодирующие чужеродные белки; агрономические гены; гены устойчивости к заболеваниям растений; гены, придающие устойчивость к паразитам растений; гены, придающие устойчивость к гербицидам; и гены, которые придают или вносят вклад в признак, являющийся положительным дополнением (например, модифицированный метаболизм жирных кислот; сниженное содержание фитатов; и модифицированный углеводный состав). Примеры всех из вышеупомянутых последовательностей нуклеиновых кислот известны специалистам в данной области.
Ген Rf4 также можно вводить в организм с одним или несколькими маркерными генами, функционально связанными с регуляторным элементом (например, промотором), который позволяет выделение трансформированных клеток, содержащих маркер, либо с помощью отрицательной селекции (т.е. ингибирования роста клеток, которые не содержат ген селективного маркера), либо с помощью положительной селекции (т.е. скрининга на продукт, кодируемый генетическим маркером). Многие гены селективных маркеров для трансформации хорошо известны в данной области и включают, например, гены, которые кодируют ферменты, которые осуществляют метаболическую детоксикацию селективного химического агента, который может представлять собой антибиотик или гербицид, или гены, которые кодируют измененную мишень, которая может быть нечувствительной к ингибитору. В данной области также известно Несколько способов положительной селекции. Примеры маркерных генов, пригодных для применения в клетках растений, могут включать, например и не ограничиваясь ими: ген неомицинфосфотрансферазы II (nptII) (Fraley et al. (1983) Proc. Natl. Acad. Sci. USA 80:4803); ген гигромицинфосфотрансферазы (Vanden Elzen et al. (1985) Plant Mol. Biol. 5:299); ген гентамицинацетилтрансферазы, ген стрептомицинфосфотрансферазы, аминогликозид-3'-аденилтрансферазы, и ген, определяющий устойчивость к блеомицину (см., например, Hayford et al. (1988) Plant Physiol. 86:1216; Jones et al. (1987) Mol. Gen. Genet. 210:86); Svab et al. (1990) Plant Mol. Biol. 14:197; и Hille et al. (1986) Plant Mol. Biol. 7:171); гены селективных маркеров, которые придают устойчивость к гербицидам, таким как глифосат, глюфосинат или бромксинил (см., например, Comai et al. (1985) Nature 317:741-744; Gordon-Kamm et al. (1990) Plant Cell 2:603-618; и Stalker et al. (1988) Science 242:419-423); ген дигидрофолатредуктазы мыши (Eichholtz et al. (1987) Somatic Cell Mol. Genet. 13:67); ген 5-енолпирувилшикимат-3-фосфатсинтетазы растений (Shah et al. (1986) Science 233:478); ген ацетолактатсинтетазы растений (Charest et al. (1990) Plant Cell Rep. 8:643).
Для другого класса маркерных генов, пригодных для трансформации растений, используется скрининг предположительно трансформированных клеток-растений, а не прямая генетическая селекция трансформированных клеток в отношении устойчивости к токсическому веществу, такому как антибиотик. Эти гены особенно пригодны для количественного определения или визуализации пространственного характера экспрессии гена в конкретных тканях, и их часто называют "репортерными генами", поскольку они могут быть слиты с геном или регуляторной последовательностью гена для исследования экспрессии гена. Широко используемые гены для скрининга трансформированных клеток включают ген β-глюкуронидазы (GUS), β-галактозидазы, люциферазы и хлорамфениколацетилтрансферазы. См., например, Jefferson (1987) Plant Mol. Biol. Rep. 5:387; Teeri et al. (1989) EMBO J. 8:343; Koncz et al. (1987) Proc. Natl. Acad. Sci U.S.A. 84:131; и DeBlock et al. (1984) EMBO J. 3: 1681.
Недавно стали более доступными способы для визуализации активности GUS in vivo, которые не требуют разрушения ткани растений. Molecular Probes publication 2908, Imagene Green.TM., p. 1-4, 1993; и Naleway et al. (1991) J. Cell Biol. 115: 151a. Кроме того, гены, кодирующие флуоресцентные белки (например, GFP, EGFP, EBFP, ECFP и YFP) используют в качестве маркеров для экспрессии генов в прокариотических и эукариотических клетках. См. Chalfie et al. (1994) Science 263:802. В качестве маркеров для скрининга можно использовать флуоресцентные белки и мутации флуоресцентных белков.
В некоторых вариантах осуществления ген Rf4 кукурузы и фрагменты или сегменты гена Rf4 кукурузы, описанные в настоящем описании, можно использовать для идентификации гомологичных последовательностей Rf4 из организмов, отличных от кукурузы (например, путем сравнения последовательностей). Последовательности из организмов, отличных от кукурузы, которые являются гомологичными гену Rf4 кукурузы, можно идентифицировать и выделять хорошо известными способами, например, на основе их гомологии последовательности с Rf4-bHLH. Например, всю кодирующую последовательность Rf4-bHLH или ее часть можно использовать в качестве зонда, который специфично гибридизуется с другими последовательностями, присутствующими в совокупности клонированных фрагментов геномной ДНК (т.е. геномной библиотеке) из организма в соответствии со стандартными способами. Таким образом, в некоторых вариантах осуществления изобретение включает нуклеотидные последовательности, которые специфично гибридизуются с последовательностью Rf4-bHLH (например, SEQ ID NO: 220).
Альтернативно последовательности из организмов, отличных от кукурузы, которые гомологичны гену Rf4 кукурузы, можно идентифицировать и выделять путем сравнения последовательностей. Например, можно проводить поиск в полностью или частично отсеквенированном геноме организма в соответствии со стандартными способами с помощью последовательности Rf4-bHLH кукурузы (например, SEQ ID NO: 220) для идентификации генов в геноме организма, которые обладают высокой степенью идентичностью последовательности с Rf4 кукурузы, и, таким образом, вероятно являются гомологами Rf4.
Например, всю последовательность Rf4 кукурузы или ее часть (например, SEQ ID NO: 220) можно использовать в качестве "эталонной последовательности". Как правило, последовательности нуклеиновых кислот (например, клонированные или геномные фрагменты ДНК геномной библиотеки), которые сравнивают с эталонной последовательностью, включают "окно сравнения", которое представляет собой определенный непрерывный сегмент последовательности нуклеиновой кислоты. Окно сравнения может включать вставки или делеции (т.е. пропуски) по сравнению с эталонной последовательностью (которая не содержит вставок или делеций) для оптимального выравнивания двух последовательностей. Окно сравнения, как правило, имеет длину по меньшей мере 20 последовательно расположенных нуклеотидов, однако оно может иметь длину 30, 40, 50, 100 или 200 нуклеотидов или более. Для избежания высокого сходства с эталонной последовательностью вследствие включения делеций в окно сравнения полинуклеотидных последовательностей, может быть внесен "штраф за делецию", чтобы его вычитать из количества соответствующих друг другу нуклеотидов.
Способы выравнивания последовательностей для сравнения хорошо известны в данной области. Определение процентной идентичности последовательностей между любыми двумя последовательностями можно проводить с использованием доступных математических алгоритмов. Неограничивающими примерами таких математических алгоритмов являются алгоритм Myers and Miller (1988), CABIOS 4:11-7; алгоритм локального выравнивания Smith et al. (1981) Adv. Appl. Math. 2:482; алгоритм общего выравнивания Needleman and Wunsch (1970), J. Mol. Biol. 48:443-53; способ поиска для локального выравнивания Pearson and Lipman (1988), Proc. Natl. Acad. Sci. USA 85:2444-8; алгоритм Karlin and Altschul (1990), Proc. Natl. Acad. Sci. USA 87:2264, и Karlin and Altschul (1993) Proc. Natl. Acad. Sci. USA 90:5873-7.
Специалист в данной области может осуществлять эти математические алгоритмы на компьютере для сравнения последовательностей в целях определения идентичности последовательностей, или в целях поиска в базе данных, содержащей множество последовательностей (например, база данных геномов организмов) в зависимости от идентичности последовательности с эталонной последовательностью. Такие осуществления включают, но не ограничиваются ими, CLUSTAL в программе PC/Gene (Intelligenetics, Mountain View, CA); и программу ALIGN и GAP, BESTFIT, BLAST, FASTA и TFASTA в GCG Wisconsin Genetics Software Package, v.10 (Accelrys Inc., San Diego, CA). Выравнивание последовательностей с использованием этих программ можно проводить, используя их параметры по умолчанию. Альтернативно может быть желательной модификация параметров по умолчанию при некоторых поисках (например, изменение величины штрафа за делецию). Выбор конкретного компьютерного осуществления математических алгоритмов для вычисления идентичности последовательностей и выбор величин параметров для применения в выбранном алгоритме находятся на усмотрении специалиста в данной области.
В некоторых вариантах осуществления систему CMS-C/Rf4 для получения гибридных семян можно включать способами инженерии в сорт кукурузы, лишенный функционального гена-восстановителя Rf4, или вид растений, отличный от кукурузы, например, путем введения гена Rf4 в такой сорт кукурузы или вид растений.
Таким образом, в соответствии с некоторыми вариантами осуществления, ген Rf4, описанный в настоящем описании, можно использовать в способе получения гибридных семян. Способ получения гибридных семян может включать получение молекулы нуклеиновой кислоты, содержащей последовательность Rf4-bHLH кукурузы (например, SEQ ID NO: 220), или нуклеотидную последовательность, которая специфично гибридизуется с последовательностью Rf4-bHLH кукурузы. Затем эту молекулу нуклеиновой кислоты можно вводить в растительную клетку или растительную ткань, где растение, из которого получена растительная клетка или ткань растений, может представлять собой Zea mays, или другой вид растений. Затем из растительной клетки или ткани растений можно получать трансформированное целое растение, в которое введена молекула нуклеиновой кислоты. Затем растение с цитоплазматической мужской стерильностью можно опылять с помощью трансформированного цельного растения. Затем из растения с цитоплазматической мужской стерильностью можно получать семена, которые были опылены трансформированным цельным растением.
В конкретных вариантах осуществления функциональные варианты или гомологи гена Rf4-bHLH кукурузы можно использовать вместо последовательности Rf4-bHLH кукурузы (например, SEQ ID NO: 220), или нуклеотидной последовательности, которая специфично гибридизуется с последовательностью Rf4-bHLH кукурузы, в способе получения гибридных семян. Трансформированное цельное растение, которое получают, например, описанными выше способами, может быть способно продуцировать семена. Однако такие семена могут быть неспособны вырастать в фертильные растения. Таким образом, некоторые варианты осуществления способов получения гибридных семян вовлекают способы культивирования тканей растений. Такие способы являются стандартными и широко известны специалистам в данной области.
В вариантах осуществления, где систему CMS-C/Rf4 для получения гибридных семян вводят способами инженерии в виды растений, отличные от кукурузы, может быть необходимо также вводить молекулы нуклеиновых кислот, содержащие одну или несколько последовательность(ей) нуклеиновых кислот, вовлеченных в систему мужской стерильности CMS-C, в виды растений. Например, рецессивный аллель rf4-bHLH можно вводить для замены ортолога Rf4-bHLH в виде растений для получения растения с мужской стерильностью rf4/rf4, в которое можно вводить ген Rf4-bHLH для создания в виде способами инженерии системы CMS-C/Rf4 для получения гибридных семян.
В некоторых вариантах осуществления ген Rf4, описанный в настоящем описании, можно использовать в способе получения гибридных семян, включающем оплодотворение женского растения, имеющего признак мужской стерильности CMS C-типа пыльцой из мужского растения, содержащего ген Rf4. В этих и других вариантах осуществления можно использовать последовательность Rf4-bHLH кукурузы (например, SEQ ID NO: 220), нуклеотидную последовательность, которая специфично гибридизуется с последовательностью Rf4-bHLH кукурузы, или функциональные варианты или гомологи последовательности Rf4-bHH.
В некоторых вариантах осуществления способ получения гибридных семян включает получение первого растения, содержащего Rf4, например, путем обратного скрещивания; мутагенеза; трансформации; или гомологичной рекомбинации. Затем можно получать второе растение, имеющее признак мужской стерильности CMS C-типа, или создавать, например, путем обратного скрещивания; мутагенеза; или гомологичной рекомбинации. Затем второе растение можно скрещивать с первым растением с получением фертильных гибридных семян из второго растения. В вариантах осуществления первое растение может представлять собой мужское растение, а второе растение может представлять собой женское растение.
В конкретных примерах способов получения гибридных семян растение может представлять собой растение кукурузы. В следующих примерах можно использовать растения, отличные от кукурузы. Варианты осуществления способов получения гибридных семян в соответствии с изобретением применимы к любому растению, такому как растения, размножающиеся половым путем, включая растения, имеющие агрономическую ценность, например и не ограничиваясь ими: кукурузу; сою; люцерну; пшеницу; рапс; рис; сорго; сахарную свеклу; брахиподию; однодольные растения; двудольные растения; различные овощи, включая огурец, помидор, перец и т.д.; различные деревья, включая яблоко, грушу, персик, вишню, красное дерево, сосну, дуб и т.д.; и различные декоративные растения.
V. Способы применения молекулярных маркеров Rf4
Способы применения молекулярных маркеров нуклеиновых кислот, которые сцеплены с геном Rf4 или расположены в нем, для идентификации растений с функциональным геном-восстановителем для CMS C-типа могут приводить к экономии затрат для растениеводов, поскольку такие способы могут устранить потребность в скрещивании растений, содержащих функциональный ген-восстановитель, с линиями растений CMS, а затем фенотипировании потомства скрещивания.
Дополнительные маркеры можно идентифицировать в качестве эквивалента любому из иллюстративных маркеров, названных в настоящем описании (например, SEQ ID NO: 1-197 и полиморфизмы ID №№ 1-106 (таблица 3)), например, путем определения частоты рекомбинации между дополнительным маркером и иллюстративным названным маркером. При таком определении можно использовать усовершенствованный способ ортогональных контрастов на основе способа Mather (1931), The Measurement of Linkage in Heredity, Methuen & Co., London, с последующим тестом на максимальную вероятность для определения частоты рекомбинации. Allard (1956) Hilgardia 24:235-78. Если величина частоты рекомбинации меньше или равна 0,10 (т.е. 10%) в любом культиваре кукурузы, то дополнительный маркер считается эквивалентом конкретному эталонному маркеру для целей использования в настоящих описанных способах.
Средства для восстановления фертильности кукурузы CMS-C могут включать последовательность нуклеиновой кислоты из растения, выявление которой обеспечивает по меньшей мере убедительное указание на то, что растение, содержащее последовательность нуклеиновой кислоты, содержит функциональный восстановитель гена CMS-C. В некоторых примерах средство для восстановления фертильности кукурузы CMS-C представляет собой маркер, который сцеплен (например, сцеплен; прочно сцеплен; или чрезвычайно прочно сцеплен) с геном Rf4-bHLH или который расположен в нем.
Средства для идентификации растений кукурузы, имеющих ген для восстановления фертильности кукурузы CMS-C, может представлять собой молекулу, которая обеспечивает поддающийся детекции сигнал при добавлении к образцу, полученному из растения, имеющего ген для восстановления фертильности кукурузы CMS-C. Специфическая гибридизация нуклеиновых кислот представляет собой поддающийся детекции сигнал, и, таким образом, зонд нуклеиновой кислоты, который специфично гибридизуется с репортерным геном CMS-C, или отличающейся геномной последовательностью нуклеиновой кислоты, которая является индикатором присутствия функционального гена-восстановителя CMS-C, может быть средством для идентификации растений кукурузы, имеющих ген для восстановления фертильности кукурузы CMS-C. В некоторых примерах средства для идентификации растений, имеющих ген для восстановления фертильности кукурузы CMS-C, представляют собой зонд, который специфично гибридизуется с маркером, который сцеплен (например, сцеплен; прочно сцеплен; или чрезвычайно прочно сцеплен) с геном Rf4-bHLH кукурузы или который расположен в нем.
В некоторых вариантах осуществления маркеры, фланкирующие ген Rf4, можно использовать для переноса сегмента(ов) донорной родительской ДНК, которая однозначно содержит ген Rf4. В конкретных вариантах осуществления маркеры выбирают из группы маркеров, содержащей SEQ ID NO: 1-197 и полиморфизмы ID №№ 1-106 (таблица 3), или из маркеров, эквивалентных маркерам, выбранным из группы маркеров, содержащей SEQ ID NO: 1-197 и полиморфизмы ID №№ 1-106 (таблица 3). В некоторых вариантах осуществления способ использования маркеров, фланкирующих ген Rf4, для переноса сегмента(ов) донорной родительской ДНК, которая однозначно содержит ген Rf4, может включать анализ геномной ДНК двух родительских растений с помощью зондов, которые специфично гибридизуются с маркерами, сцепленными (например, сцепленными; прочно сцепленными; или чрезвычайно прочно сцепленными) с геном Rf4; половое скрещивание двух генотипов родительских растений с получением популяции потомков, и анализ этих потомков на присутствие маркеров, сцепленных (например, сцепленных; прочно сцепленных; или чрезвычайно прочно сцепленных) с геном Rf4; обратное скрещивание потомков, которые содержат маркеры, сцепленные (например, сцепленные; прочно сцепленные; или чрезвычайно прочно сцепленные) с геном Rf4, с реципиентным генотипом для получения популяции первого возвратного скрещивания, а затем продолжение программы обратного скрещивания до получения конечного потомства, которое содержит любой желаемый признак(и), проявляемый родительским генотипом и геном Rf4. В конкретных вариантах осуществления селекцию индивидуального потомства, полученного на каждой стадии скрещивания и обратного скрещивания, проводят с помощью анализа маркера Rf4 в каждом поколении. В некоторых вариантах осуществления анализ геномной ДНК двух родительских растений с помощью зондов, которые специфично гибридизуются с маркерами, сцепленными (например, сцепленными; прочно сцепленными; или чрезвычайно прочно сцепленными) с геном Rf4 показывает, что одно из родительских растений содержит меньше сцепленных маркеров, с которыми зонды специфично гибридизуются, или ни один из сцепленных маркеров, с которыми зонды специфично гибридизуются.
В некоторых вариантах осуществления маркеры, которые сцеплены (например, сцеплены; прочно сцеплены; или чрезвычайно прочно сцеплены) с геном Rf4-bHLH кукурузы или которые расположены в нем, или саму последовательность гена Rf4-bHLH кукурузы можно использовать для введения гена Rf4 кукурузы в растение путем генетической трансформации. В конкретных вариантах осуществления маркеры выбраны из группы маркеров, содержащих SEQ ID NO: 1-197 и полиморфизм ID №№ 1-106 (таблица 3), или из маркеров, эквивалентных маркерам, выбранным из группы маркеров, содержащей SEQ ID NO: 1-197 и полиморфизмы ID №№ 1-106 (таблица 3). В некоторых вариантах осуществления способ введения гена Rf4 кукурузы в растение кукурузы путем генетической рекомбинации может включать анализ геномной ДНК растения (например, растения кукурузы) с помощью зондов, которые специфично гибридизуются с маркерами, сцепленными (например, сцепленными; прочно сцепленными; или чрезвычайно прочно сцепленными) с геном Rf4, или с самим геном Rf4 для идентификации гена Rf4 в растении; выделение сегмента геномной ДНК растения, содержащего ген Rf4, например, путем экстракции геномной ДНК и расщепления геномной ДНК одним или несколькими ферментами - эндонуклеазами рестрикции; необязательно амплификацию выделенного сегмента ДНК; введение выделенного сегмента ДНК в клетку или ткань хозяйского растения кукурузы; и анализ ДНК хозяйского растения кукурузы с помощью зондов, которые специфично гибридизуются с маркерами, сцепленными (например, сцепленными; прочно сцепленными; или чрезвычайно прочно сцепленными) с геном Rf4, или с самим геном Rf4, для идентификации гена Rf4 в хозяйском растении кукурузы. В конкретных вариантах осуществления выделенный сегмент ДНК можно вводить в хозяйское растение кукурузы, так чтобы он стабильно встраивался в геном хозяйского растения кукурузы.
В некоторых вариантах осуществления маркеры, которые сцеплены (например, сцеплены; прочно сцеплены; или чрезвычайно прочно сцеплены) или с геном Rf4-bHLH кукурузы или расположены в нем, или саму последовательность гена Rf4-bHLH кукурузы можно использовать для введения гена Rf4 кукурузы в другие организмы, например, растения. В конкретных вариантах осуществления маркеры выбирают из группы маркеров, включающей SEQ ID NO: 1-197 и полиморфизмы ID №№ 1-106 (таблица 3), или из маркеров, эквивалентных маркерам, выбранным из группы маркеров, содержащей SEQ ID NO: 1-197 и полиморфизмы ID №№ 1-106 (таблица 3). В некоторых вариантах осуществления способ введения гена Rf4 кукурузы в организм, отличный от кукурузы, может включать анализ геномной ДНК растения (например, растения кукурузы) с помощью зондов, которые специфично гибридизуются с маркерами, сцепленными (например, сцепленными; прочно сцепленными; или чрезвычайно прочно сцепленными) с геном Rf4, или с самим геном Rf4, для идентификации гена Rf4 в растении; выделение сегмента геномной ДНК растения, содержащей ген Rf4, например, путем экстракции геномной ДНК и расщепления геномной ДНК одним или несколькими ферментами - эндонуклеазами рестрикции; необязательно амплификацию выделенного сегмента ДНК; введения выделенного сегмента ДНК в организм, отличный от кукурузы; и анализ ДНК организма, отличного от кукурузы, с помощью зондов, которые специфично гибридизуются с маркерами, сцепленными (например, сцеплены; прочно сцеплены; или чрезвычайно прочно сцеплены) с геном Rf4, или с самим геном Rf4 для идентификации гена Rf4 в организме. В конкретных вариантах осуществления выделенный сегмент ДНК можно вводить в организм, так чтобы он стабильно встраивался в геном организма.
В некоторых вариантах осуществления маркеры, которые сцеплены (например, сцеплены; прочно сцеплены; или чрезвычайно прочно сцеплены) или которые расположены в гене Rf4, или саму последовательность гена Rf4 можно использовать для идентификации растения с функциональным геном-восстановителем мужской стерильности CMS-C. В конкретных вариантах осуществления растение представляет собой растение кукурузы. В некоторых вариантах осуществления молекулы нуклеиновых кислот (например, геномную ДНК или мРНК) можно экстрагировать из растения. Затем экстрагированные молекулы нуклеиновых кислот можно контактировать с одним или несколькими зондами, которые специфично гибридизуются с маркерами, сцепленными (например, сцепленными; прочно сцепленными; или чрезвычайно прочно сцепленными) с геном Rf4, или с самим геном Rf4. Специфическая гибридизация одного или нескольких зондов с экстрагированными молекулами нуклеиновых кислот указывает на присутствие функционального гена-восстановителя для мужской стерильности CMS-C в растении.
В некоторых вариантах осуществления маркеры, которые сцеплены (например, сцеплены; прочно сцеплены; или чрезвычайно прочно сцеплены) с геном Rf4 или которые расположены в нем, или саму последовательность гена Rf4 можно использовать для получения гибридных семян. Получение гибридных семян в соответствии с такими способами может приводить к экономии затрат благодаря устранению ручного или механического удаления метелок, и, кроме того, может увеличить выход семян. В конкретных вариантах осуществления способ может включать скрещивание мужского растения, содержащего молекулярные маркеры нуклеиновых кислот, которые сцеплены (например, сцеплены; прочно сцеплены; или чрезвычайно прочно сцеплены) или с геном Rf4 или расположены в нем, и женского растения, имеющего фенотип мужской стерильности CMS C-типа.
VI. Организмы, содержащие ген Rf4
Некоторые варианты осуществления настоящего изобретения также относятся к организму, включающему молекулу нуклеиновой кислоты, содержащую последовательность Rf4-bHLH (например, SEQ ID NO: 220), последовательность нуклеиновой кислоты, которая специфично гибридизуется с последовательностью Rf4-bHLH, или функциональный вариант последовательности Rf4-bHLH. Подходящий организм может представлять собой любое подходящее растение, дрожжи или бактерию. В качестве неограничивающего примера, растение, содержащее упомянутые выше последовательности, может представлять собой растение, представляющее агрономическую ценность, например и не ограничиваясь ими: кукурузу; сою; люцерну; пшеницу; рапс; рис; сорго; свеклу; брахиподию; однодольные растения; двудольные растения; различные овощи, включая огурец, помидор, перец и т.д.; различные деревья, включающие яблоко, грушу, персик, вишню, красное дерево, сосну, дуб и т.д.; и различные декоративные растения. В конкретных вариантах осуществления организм может представлять собой растение, размножающееся половым путем. Несущее семена растение, которое содержит конкретную последовательность нуклеиновой кислоты, может продуцировать семена, которые содержат эту последовательность нуклеиновой кислоты.
Клетки растений, содержащие последовательность Rf4-bHLH (например, SEQ ID NO: 220), последовательность нуклеиновой кислоты, которая специфично гибридизуется с последовательностью Rf4-bHLH, или функциональным вариантом последовательности Rf4-bHLH, можно культивировать и поддерживать в качестве клеток культуры ткани растений, или в культуральную среду можно добавлять определенные растительные гормоны, известные в данной области, тем самым обеспечивая дифференцировку клеток культуры ткани растений и формирование нового сорта растений, который может быть фертильным или стерильным. Такие способы культивирования растений, пригодные в этих и других вариантах осуществления, являются стандартными и хорошо известны в данной области.
Некоторые варианты осуществления изобретения относятся к вирусу (например, бактериофагу или вирусу растений), содержащему последовательность Rf4-bHLH (например, SEQ ID NO: 220), последовательность нуклеиновой кислоты, которая специфично гибридизуется с последовательностью Rf4-bHLH, или функциональный вариант последовательности Rf4-bHLH.
Следующие примеры предоставлены для иллюстрации определенных конкретных признаков и/или вариантов осуществления. Примеры не следует истолковывать, как ограничивающие изобретение до конкретных проиллюстрированных признаков или вариантов осуществления.
ПРИМЕРЫ
Пример 1: Материалы и способы
Популяция для подтверждения
Линию мужской стерильности CMS C-типа, BE4207, и линию-восстановитель мужской стерильности, соответствующей CMS C-типа, XJH58, использовали в качестве родительских растений для получения потомства F1. Затем потомство F1 подвергали самоопылению для получения популяции F2. Популяцию F2, состоящую из 500 особей, использовали для идентификации гена Rf4 и маркеров, сцепленных (например, сцепленных; прочно сцепленных; или чрезвычайно прочно сцепленных) с геном Rf4.
Точное картирование популяции F3 BE4207/XJH58
Всего 5465 семян, отобранных из 15 гетерозиготных семейств F2 из популяции для подтверждения F2, сегрегирующей по различным фрагментам в области размером 4,2 млн.п.н. в верхней части 8 хромосомы, высевали в рассаднике летом 2010 года в Arlington, Wisconsin. Образцы листьев собирали от 5104 проросших сеянцев для генотипирования.
Классификация по фертильности
500 растений в этой популяции F2 фенотипически классифицировали в зависимости от пыльцы, сбрасываемой с метелок. Растения, которые сбрасывали пыльцу, классифицировали как фертильные. Растения, которые не сбрасывали пыльцу, классифицировали как стерильные. Восстановление Rf4 в этой популяции было полным; не наблюдали частично фертильных растений.
Экстракция и количественное определение ДНК
8 полученных прокалыванием дыроколом фрагментов ткани листьев получали от каждого растения популяции F2, и ДНК экстрагировали с использованием Biocel™ 1800 (Agilent Inc., Santa Clara, CA). Использованный способ экстракции ДНК был следующим: (1) добавить одну гранулу из сплава вольфрама диаметром ~0,32 см в каждую пробирку; (2) добавить 300 мкл лизирующего буфера RLT (Qiagen Inc., Germantown, MD) в каждую пробирку; (3) закрыть и размалывать в течение 6 минут при 1500 ударов/минута в SPEX 2000 Geno/Grinder® (OPS Diagnostics, LLC, Lebanon, NJ); (4) центрифугировать образцы при 6000 об/мин в течение 5 минут; (5) открыть пробирки; следующие стадии проводят на Biocel™ 1800: (6) перенести 200 мкл супернатанта в 1,1-мл планшет для анализа с квадратными лунками с круглым дном, содержащий 10 мкл MagAttract® Suspension G Beads (Qiagen Inc.); (7) инкубировать в течение 2 минут; (8) встряхивать при 1200 об/мин в течение 40 секунд; (9) инкубировать в течение 2 минут; (10) поместить планшет для анализа на магнитную полку и позволить гранулам разделиться в течение 40 секунд; (11) удалить супернатант; (12) первое промывание - добавить промывочный буфер 190 RPW™ предварительно смешанный с РНКазой и изопропанолом, и встряхивать при 1200 об/мин в течение 40 секунд; (13) поместить планшет для анализа на магнитную полку и позволить гранулам разделиться в течение 20 секунд; (14) удалить супернатант; (15) второе промывание - добавить 190 мкл, промывочного буфера, представляющего собой 100% этанол, и встряхивать при 1200 об/мин в течение 40 секунд; (16) поместить планшет для анализа на магнитную полку и позволить гранулам разделяться в течение 20 секунд; (17) третье промывание - добавить 190 мкл промывочного буфера, представляющего собой 100% этанол; (18) встряхивать при 1200 об/мин в течение 40 секунд; (19) поместить планшет для анализа на магнитную полку и позволить гранулам разделиться в течение 20 секунд; (20) удалить супернатант; (21) инкубировать планшет в течение 5 минут при комнатной температуре; (22) добавить 100 мкл буфера для элюирования AE™ (Qiagen Inc.); (23) встряхивать в течение 2 минут; (24) поместить планшет для анализа на магнитную полку и позволить гранулам разделиться в течение 30 секунд; и (25) перенести супернатант в чистый обозначенный планшет и закрыть. ДНК хранили при 4°C. ДНК количественно определяли с использованием PicoGreen® (Invitrogen Inc., Carlsbad, CA), и концентрацию доводили до 5-6 нг/мкл для применения в системе генотипирования KASPar™ (KBioscience Inc., Hoddesdon, Великобритания).
Система генотипирования SNP KASPar™
Система конкурентного аллелеспецифического генотипирования с использованием ПЦР (KASPar™) представляет собой систему для выявления SNP, в которой используется технология, основанная на аллелеспецифическом удлинении олигонуклеотидов и резонансном переносе энергии флуоресценции (FRET) для генерирования сигнала. Для каждого SNP-маркера в анализе KASPar™ требуется только два компонента: смесь для анализа (смесь трех немеченых праймеров: двух аллелеспецифических олигонуклеотидов и одного общего обратного локус-спсецифического олигонуклеотида); и реакционная смесь (другие компоненты, требуемые для ПЦР, включая универсальную флуоресцентную репортерную систему и Taq-полимеразу).
Для моделирования праймеров использовали систему KBioscience Laboratory Information Management System (KLIMS™) (KBioscience Inc.), и олигонуклеотиды синтезировали с помощью технологии Integrated DNA Technology (Coralville, IA). Реакции KASPar™ проводили в соответствии с рекомендацией изготовителя. ПЦР начинали с денатурации при 94°C в течение 15 минут, затем проводили 20 циклов по 10 секунд денатурации при 94°C, 5 секунд отжига при 57°C, а затем 10 секунд удлинения при 72°C, а за этими 20 циклами следовали 22 цикла с денатурацией в течение 10 секунд при 94°C, отжигом в течение 20 секунд при 57°C, а затем удлинением в течение 40 секунд при 72°C. Флуоресцентные сигналы после завершения реакций KASPar™ считывали в спектрофотометре (Tecan GENios™, Mannedorf, Швейцария) с длиной волны возбуждения при 485 нм, и длиной волны испускания при 535 нм для флуорофора FAM; и длиной волны возбуждения 525 нм, и длиной волны испускания 560 нм для флуорофора VIC. Данные анализировали с использованием программного обеспечения Klustercaller™ (KBiosciences Inc.) для определения генотипов каждого SNP-маркера в популяции.
Экстракция РНК и ПЦР в реальном времени (RT-ПЦР)
Родительские растения и растения F3, сегрегирующие по области Rf4, выращивали в теплице. Ткани листьев собирали с растений, выращиваемых в течение 5 недель, 7 недель и 9 недель. Также собирали ткани метелок с развивающимися пыльниками/пыльцой и сбрасываемую пыльцу (в фертильных растениях). Тотальную РНК экстрагировали с использованием набора RNeasy™ Plant Mini Kit (Qiagen Inc.). кДНК синтезировали с использованием набора QuantiTect™ Reverse Transcription Kit (Qiagen Inc.). Для RT-ПЦР, специфичные к гену Rf4 праймеры, контрольные праймеры для инвертазы кукурузы, и зонды с двойным мечением красителями FAM или VIC и Minor Groove Binding Non Fluorescence Quencher™ I (MGBNFQ) синтезировали в Applied Biosystems (Foster City, CA). Для проведения 10-мкл реакций ПЦР использовали основную смесь для генотипирования TaqMan™ (Applied Biosystems), и ПЦР проводили на LightCyclerTM 480 (Roche). Программа ПЦР включала: активацию в течение 10 минут при 95°C, затем 50 циклов при 95°C в течение 10 секунд и 58°C в течение 38 секунд. Флуоресцентные сигналы регистрировали в конце каждого цикла. Относительный уровень экспрессии вычисляли с использованием способа Delta CT с использованием инвертазы в качестве контроля.
Пример 2: Картирование гена Rf4
Анализ сегрегации фертильности
Соотношение сегрегации фертильности в популяции F2 составляло 3:1. Таблица 1. Результаты показали, что восстановление фертильности в системе CMS-C/Rf4 контролируется одним доминантным геном-восстановителем, Rf4.
Данные о фенотипе популяции для подтверждения
Предварительное генетическое картирование Rf4 в популяции F2 с использованием SNP-маркеров.
101 SNP-маркер, расположенный вблизи верхней части 8 хромосомы кукурузы, использовали в скрининге родительских организмов с 5 различными популяциями для картирования Rf4, и было определено, что он находится в пределах области Rf4 размером 5,0 млн.п.н. Набор из 12 маркеров был полиморфным во всех пяти популяциях, в то время как 27, в том числе 12 общих полиморфных маркеров, продемонстрировали полиморфизм между родительскими организмами популяции F2 BE4207×XJH58. 12 общих маркеров первоначально использовали для генотипирования всех 500 индивидуумов в популяции F2 для идентификации рекомбинантных линий в пределах области 5,0 млн.п.н. Затем остальные 15 полиморфных маркеров использовали для генотипирования всех 104 рекомбинантных линий. Тридцать четыре случайным образом отобранных рекомбинанта представлены на ФИГ.2 с их фенотипическими данными и соответствующими генотипическими данными для 27 маркеров в качестве примеров. Более детальный анализ 42 наиболее информативных рекомбинантных линий показал, что ген Rf4 расположен в области приблизительно 1,505 млн.п.н. в верхней части 8 хромосомы, определяемой SNP-маркером DAS-PZ-40624 (SEQ ID NO: 8). В этой области находится приблизительно 30 генов (данные не представлены).
Интересно и неожиданно, что, ввиду того факта, что все ранее идентифицированные гены-восстановители, за исключением Rf2 кукурузы и Rf17 риса, кодируют белки с пентатрикопептидными повторами (PPR), отсутствует предсказанный ген PPR в пределах области размером 1,505 млн.п.н., содержащей Rf4, хотя существуют три гена PPR, расположенных на расстоянии 1,509, 4,288 и 4,748 млн.п.н. соответственно. ФИГ.3.
Генетическое точное картирование Rf4 с информативными рекомбинантами и дополнительными SNP-маркерами
Для дальнейшего точного картирования расположения на хромосоме гена-восстановителя Rf4, 96 SNP-маркеров, расположенных в положении нуклеотидов от 12507 до 1504526 на 8 хромосоме, были отобраны для исследования родительских полиморфизмов. 28 SNP-маркеров были полиморфными среди двух картированных родительских растений. 93 рекомбинанта, включая некоторые потенциально информативные рекомбинанты, не включенные в предшествующий раунд скрининга, были отобраны для генотипирования с 28 маркерами. С использованием того же сравнения фенотип/генотип, которое описано выше, ген Rf4 был положительно картирован до области 0,56 млн.п.н. с использованием 19 наиболее информативных рекомбинантов, определяемых растениями S-301 и S-115 и SNP-маркером PZE-108000459 (SEQ ID NO: 134) в качестве правой границы, как показано на ФИГ.4. Исходя из генотипических и фенотипических данных растения S-378 (стерильное), ген Rf4 может быть расположен в области менее 100 т.п.н., определяемой SNP-маркером PZE-108000086 (SEQ ID NO: 105). См. ФИГ.4. Таким образом, Rf4 был картирован до области 0,56 млн.п.н., которая содержит приблизительно 14 генов, и далее был картирован до области менее 100 т.п.н., которая содержит шесть потенциальных генов. См. таблицу 2. Последовательность гена Rf4 выбрана из группы, состоящей из GRMZM2G122853 (SEQ ID NO: 198); AC187051.4_FG005 (SEQ ID NO: 199); GRMZM2G122851 (SEQ ID NO: 200); GRMZM2G122850 (SEQ ID NO: 201); GRMZM2G582028 (SEQ ID NO: 202); GRMZM2G021276 (SEQ ID NO: 203); GRMZM2G381376 (SEQ ID NO: 204); GRMZM2G081127 (SEQ ID NO: 205); GRMZM2G085111 (SEQ ID NO: 206); GRMZM2G085038 (SEQ ID NO: 207); GRMZM2G317468 (SEQ ID NO: 208); GRMZM2G328030 (SEQ ID NO: 209); GRMZM2G029450 (SEQ ID NO: 210); и GRMZM2G077212 (SEQ ID NO: 211).
Предсказанные гены в области Rf4
Эти результаты согласуются с результатами, изложенными в постере под названием "Restoration of c-type cytoplasmic male sterility in maize: Fine-mapping of Rf4", недавно представленном Kohls et al. на 2010 Maize Genetics Conference. Kohls et al., согласно утверждению, картировали ген Rf4 до области 0,5 млн.п.н. вблизи верхней части 8 хромосомы с использованием ограниченного количества маркеров. Однако генетические материалы, использованные Kohls et al., существенно отличались от генетических материалов, использованных в настоящей описываемой работе. Важно, что, Kohls et al. выявили значительный процент только полуфертильных особей Rf4, в то время как восстановление фертильности в настоящей популяции F2 было полным (не было выявлено полуфертильных особей). Кроме того, Rf4 использовали лучшее разрешение карты (<100 т.п.н. против 500 т.п.н.) для идентификации, а также для описания многих других полезных маркеров, включая маркеры, которые чрезвычайно прочно сцеплены с Rf4.
Результаты, представленные в настоящем описании, демонстрируют картирование локуса Rf4 до очень небольшого хромосомного фрагмента и идентификацию гена Rf4. В отличие от большинства генов Rf, клонированных до настоящего времени, Rf4 практически определенно не является геном PPR. Сцепленные молекулярные маркеры, описанные в настоящем описании (включая прочено сцепленные и чрезвычайно прочно сцепленные маркеры) можно использовать для облегчения селекции с помощью маркеров линий восстановителей в системе CMS-C/Rf4 и для обеспечения разработки гибридной кукурузы с использованием только этой системы или вместе с другими системами.
Пример 3: Точное картирование гена Rf4
Получали большую популяцию F3 BE4207/XJH58 из 5104 особей, происходящих из рекомбинантных линий, описанных выше, для точного картирования.
В популяции F2 Rf4 был картирован до области менее 100 т.п.н. в верхней части хромосомы 8. Однако в этой области не существует молекулярных маркеров, за исключением PZE-108000086 с SNP в нуклеотидном положении 98468. Было предпринято два подхода для идентификации полиморфизмов для дополнительных маркеров в этом интервале. В первом подходе моделировали эксперимент по улавливанию последовательностей NimbleGen™ (Roche Inc.) (Fu et al. (2010) Plant J. 62:898-909) для установления всех полиморфизмов в области приблизительно 6,0 млн.п.н. в верхней части 8 хромосомы между линией CMS BE4207 и линией восстановителя XJH58, вместе с двумя дополнительными линиями восстановителей BE9515 и MLW03. Улавливание последовательности, секвенирование уловленных мишеней и проявление SNP проводили с помощью NimbleGen™ в соответствии с рекомендованной изготовителем методикой. Совместно с экспериментами по улавливанию NimbleGen™, неповторяющиеся последовательности ДНК в области Rf4 (верхние 100 т.п.н. 8 хромосомы) устанавливали из генома B73, и конструировали ПЦР-праймеры для амплификации геномных фрагментов двух картированных родительских организмов, BE4207 и XJH58. Фрагменты ПЦР секвенировали из обоих родительских организмов и идентифицировали полиморфизмы (как SNP, так и InDel).
Исходя из результатов обоих экспериментов, было идентифицировано несколько тысяч SNP и InDel между линией CMS BE4207 и линией-восстановителем XJH58 в пределах уловленной области размером 6 млн.п.н., включая 77 SNP и 29 InDel в первых 100 т.п.н. Таблица 3. Эксперимент по амплификации с использованием ПЦР был направлен на первые 100 т.п.н., с конкретным акцентом на двух экспрессируемых в этой области генах, гене пероксидазы растений (GRMZM2G122853 (SEQ ID NO: 198)) и гене фактора транскрипции основная спираль-петля-спираль (bHLH) (GRMZM2G021276 (SEQ ID NO: 203)). Секвенирование продуктов ПЦР идентифицировало 35 SNP и 24 InDel. Таблица 3. Примечательно, что большинство изменений, идентифицированных в фрагментах ПЦР, также были найдены в проекте улавливания последовательностей (50 из 59, или 84,7%). Взятые вместе, эти два способа открыли 106 уникальных изменений (полиморфизмы ID 1-106) между двумя картированными родительскими растениями, включающих 77 SNP и 29 InDel в заданной области Rf4 размером 100 т.п.н. Таблица 3.
Исходя из положения на физической карте и контекста последовательности, 33 уникальных изменения (24 SNP и 9 InDel) использовали для конструирования праймеров для высокопроизводительного генотипирования популяций для картирования F3 с использованием анализов KASPar™ (таблица 3), по существу, как описано в примере 1.
Таблица 3
Полиморфизмы между CMS (BE4207) и линией восстановителя (XJH58) и маркерами KASPar™ для картирования Rf4. Положение нуклеотидов в B73 основано на кукурузе B73 RefGen_v1, однако v2 имеет идентичную последовательность в пределах 100 т.п.н. 8 хромосомы.
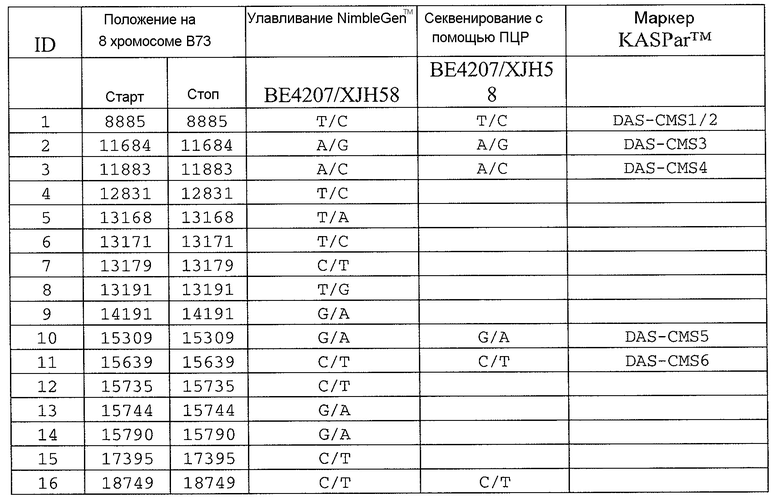
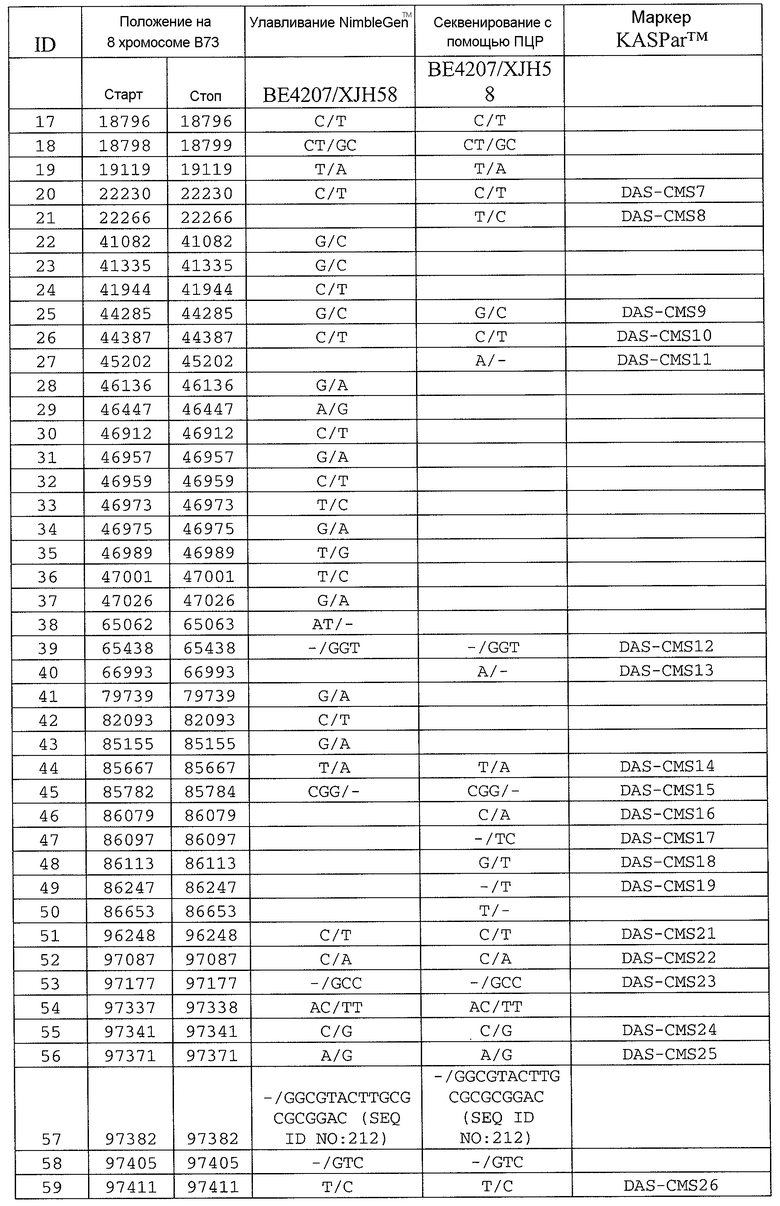
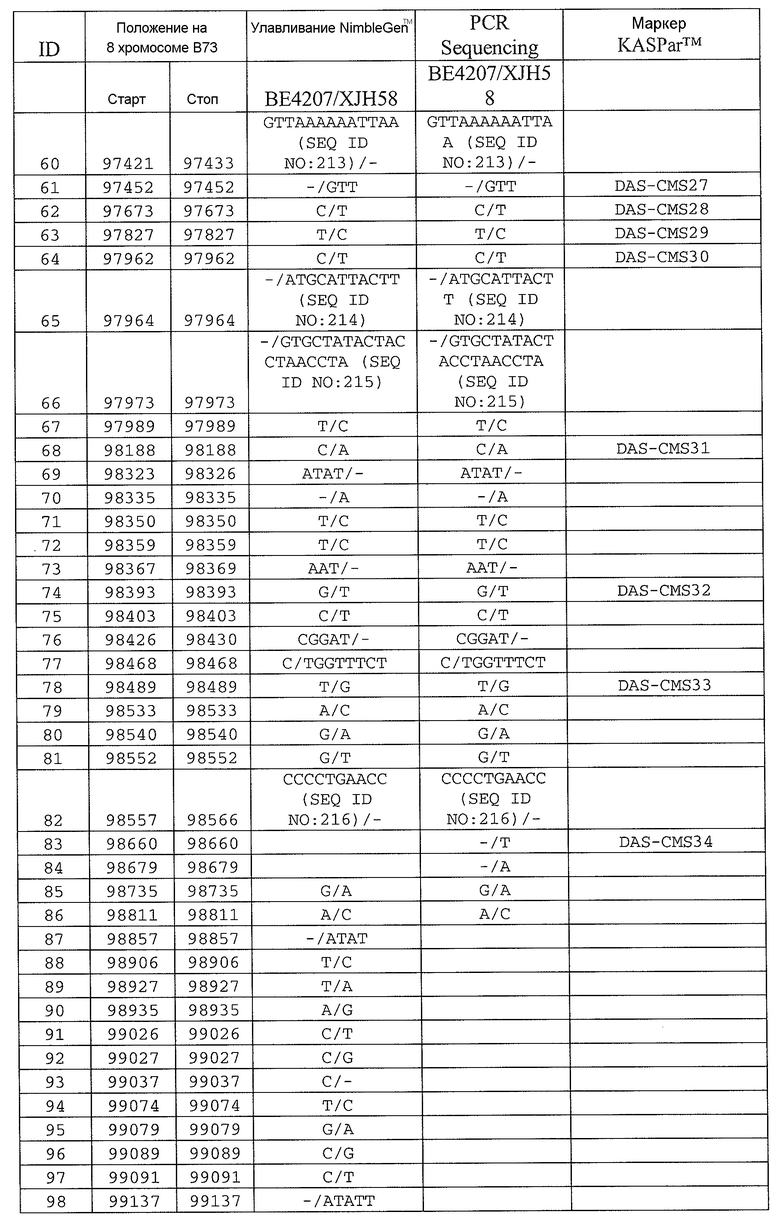
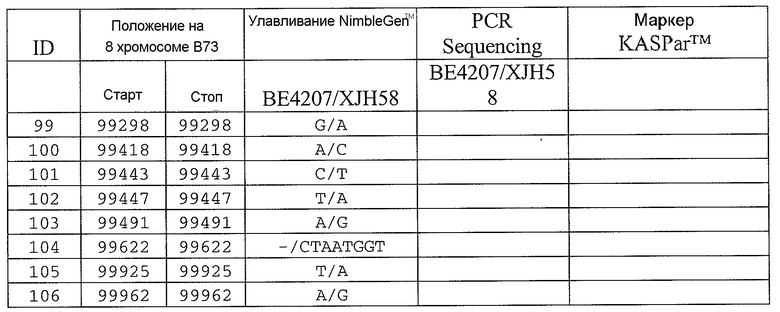
В примере 2 Rf4 был картирован до области менее 100 т.п.н. в верхней части 8 хромосомы кукурузы, определяемой SNP-маркером PZA108000086. Для картирования Rf4 до значительно меньшего интервала было выбрано четыре фланкирующих маркера (DAS-CMS1 и DAS-CMS3 слева, и PZE-108000378 и PZE-108000459 справа) для генотипирования всех 5104 растений F3 BE4207/XJH58. ФИГ.5. Растение S-378 было из F2 и являлось стерильным. ДНК из этого растения была сохранена для генотипирования с новыми маркерами. Было идентифицировано всего 307 рекомбинантов в пределах области Rf4 более 0,56 млн.п.н. Эти растения были отобраны для оценки фертильности в полевых условиях и все фертильные растения подвергали самоопылению. Кроме того, дополнительные маркеры в пределах интервала 0,56 млн.п.н. использовали для генотипирования ДНК из 307 рекомбинантов и растения S378 поколения F2, для дальнейшего определения участков рекомбинации. Точное картирование проводили путем сравнения фенотипических и генотипических данных информативных рекомбинантов. Данные 12 наиболее информативных рекомбинантов демонстрируют, что Rf4 ограничивается интервалом между DAS-CMS19 и DAS-CMS31, определяемым растениями 468-5048 и 468-4977. ФИГ.5.
С использованием генома B73 кукурузы в качестве эталона, DAS-CMS19 (-/T InDel; нуклеотидное положение 86247) и DAS-CMS31 (C/A SNP; нуклеотидное положение 98188) находятся на расстоянии приблизительно 12 т.п.н. друг от друга и располагаются в одном и том же клоне BAC; ZMMBBb0329M04 (номер доступа № AC187051). Последовательность интервала между DAS-CMS19 и DAS-CMS31 (в обоих B73 RefSeq v1 и v2) представлена в списке последовательностей в качестве SEQ ID NO: 217. Поскольку самая последняя версия B73 RefGen (v2) все еще имеет два непроясненных пропуска (нуклеотиды 88058-88157 и 94849-94948) между двумя маркерам, точная длина неизвестна. Однако эти пропуски, более вероятно, являются результатом трудности сборки вследствие повторяющихся последовательностей, а не истинными физическими пропусками, поскольку оба маркера располагаются в одном и том же клоне BAC, который отсеквенирован.
Пример 4: Охарактеризация гена Rf4 кукурузы
Rf4 кодирует фактор транскрипции bHLH
В геноме B73 интервал размером 12 т.п.н. между DAS-CMS19 и DAS-CMS31 содержит два предсказанных гена: GRMZM2G582028 (SEQ ID NO: 202) (нуклеотиды 93160-93850) и GRMZM2G021276 (SEQ ID NO: 203) (нуклеотиды 95823-98367). Первый фрагмент размером 9,4 т.п.н. (нуклеотиды 86247-95642) интервала размером 12 т.п.н. по большей части состоит из повторяющихся последовательностей, включающих GRMZM2G582028. GRMZM2G582028 аннотирован как новый перемещающийся элемент в B73 RefGen v2, и предсказано, что он кодирует небольшой белок из 68 аминокислот, который не имеет каких-либо значительных соответствий в базах данных белков. Более того, сама последовательность GRMZM2G582028 является в высокой степени повторяющейся, и множество идентичных или практически идентичных копий встречаются на всех 10 хромосомах кукурузы. Таким образом, маловероятно, чтобы GRMZM2G582028 был Rf4.
Оставшимся кандидатом для Rf4 является только другой предсказанный ген в этой области GRMZM2G021276. Этот ген кодирует фактор транскрипции основная спираль-петля-спираль (bHLH) из 365 аминокислот с консервативным доменом bHLH, расположенным приблизительно в средней части белка. Промотор/5'-UTR и полная кодирующая область, а также 3'-UTR размером 82 п.н., этого гена расположены в пределах интервала 12 т.п.н. с правым пограничным маркером (DAS-CMS31), находящимся в области 3'-UTR. Результаты BLAST указывают на то, что GRMZM2G021276 (в дальнейшем в настоящем описании обозначаемый как Rf4-bHLH) наиболее вероятно является однокопийным геном в геноме B73. Этот ген и его продукт ранее не были охаратеризованы.
Аллельные варианты Rf4-bHLH
Геномная последовательность B73 указывает на то, что ген Rf4-bHLH имеет 4 экзона и три интрона (ФИГ.6), включая интрон 1 размером 635 т.п.н., расположенный в 5'-UTR. Исходя из этой информации, моделировали ПЦР-праймеры для амплификации локуса Rf4-bHLH размером 3,2 т.п.н. из линии CMS BE4207, и трех линий восстановителей (XJH58, BE9515 и MLW03), а также из не являющегося CMS инбредного растения B104.
Результаты секвенирования указывают на то, что существуют существенное варьирование аллелей между различными инбредными растениями. ФИГ.7. Аллель-невосстановитель rf4-bHLH из BE4207 (CMS; не имеет функционального гена-восстановителя) идентичен аллелю B73, хорошо известного инбредного растения, которое не восстанавливает CMS-C. Интересно, что три известных линии восстановителей (XJH58, BE9515 и MLW03) имеют идентичный аллель (восстановитель) Rf4-bHLH, который отличается от аллеля B73/BE4207 (CMS). Линии восстановителей XJH58 и MLW03 обладают одним общим предком 2-3 поколения назад. Однако инбредное растение BE9515, которое не обладает общими предками с XJH58 или MLW03, также имеет идентичную последовательность Rf4-bHLH, указывая на то, что этот конкретный аллель, возможно, является консервативным среди линий, которые восстанавливают цитоплазму CMS-C. Между аллелем-восстановителем (Rf4-bHLH) и аллелем-невосстановителем CMS/B73 (rf4-bHLH) имеется 20 SNP и 16 InDel. ФИГ.7. Примечательно, что существует только один SNP C/T между аллелем CMS/B73 и аллелем-восстановителем в 5'-UTR/промоторной области размером 1,1 т.п.н. Когда сравнивают предсказанные кДНК из 6 аллелей, между аллелем CMS/B73 и аллелем-восстановителем имеется только 6 SNP и 2 InDel. ФИГ.8.
B104 представляет собой инбредную линию, которая не имеет цитоплазму CMS-C. Проводятся эксперименты для определения того, является ли B104 восстановителем фертильности CMS-C. Из 44 варьирований последовательности, выявленных в аллеле B104, 28 из них имеют ту же последовательность, что и аллель CMS/B73, 8 являются такими же, как и у гена-восстановителя, и 8 являются уникальными для B104 (ФИГ.7), указывая на то, что аллель B104 более сходен с аллелем CMS/B73. Интересны, что все из вариантов кДНК в B104 имеют ту же последовательность, что и аллель CMS/B73, за исключением уникальной делеции размером 12 п.н. (12 Cs), встречающейся только в 5'-UTR B104. ФИГ.8.
Выравнивание предсказанных белковых последовательностей Rf4-bHLH представлено на ФИГ.9. Как и ожидалось, все три линии-восстановителя имеют идентичные белковые последовательности, в то время как B73, BE4207 и B104 обладают идентичными белковыми последовательностями. Между двумя вариантами имеется только четыре аминокислотных изменения в линиях-восстановителях: замена His на Asn (H103 на N103; полиморфизм ID 52; DAS-CMS22), инсерция Ala (A130; полиморфизм ID 53; DAS-CMS23), замена Pro на Leu (P266 на L267; полиморфизм ID 62; DAS-CMS28), и замена Tyr на Phe (Y186 на F187; полиморфизм ID 54). Только замена Tyr на Phe встречается в консервативном домене bHLH. ФИГ.9.
Rf4-bHLH специфически экспрессируется в развивающихся метелках/пыльце
Профиль экспрессии Rf4-bHLH исследовали для получения дальнейших доказательств того, что этот фактор транскрипции участвует в восстановлении фертильности CMS-C. Тотальную РНК экстрагировали из двух картированных родительских организмов, BE4207 и XJH58, а также особей F3, происходящих из початка F2, сегрегирующего по первой области размером 0,56 млн.п.н. 8 хромосомы. Специфичные для Rf4-bHLH олигонуклеотиды и контрольные олигонуклеотиды инфертазы (праймеры и зонды), смоделированные для RT-ПЦР, представлены в таблице 4.
Олигонуклеотиды для RT-ПЦР
Данные количественной ПЦР в реальном времени демонстрируют, что в родительских растениях-восстановителях XJH58, и в растениях F3, гомозиготных или гетерозиготных по аллелю-восстановителю, Rf4-bHLH демонстрирует наиболее высокую экспрессию в метелках с развивающимися пыльниками/пыльцой, слабую экспрессию в сбрасываемой пыльце и практически отсутствие экспрессии в листьях различных стадий развития. Эти профили экспрессии согласуются с ролью Rf4-bHLH в восстановлении фертильности пыльцы.
Следовательно, rf4-bHLH демонстрирует от очень низкой до отсутствия экспрессии в листьях, а также в развивающихся метелках, в родительском организме CMS BE4207, и в растениях F3, гомозиготных по аллелю-невосстановителю BE4207. ФИГ.10. Однако отсутствие экспрессии rf4-bHLH в развивающихся метелках растений, гомозиготных по аллелю-невосстановителю BE4207, наиболее вероятно, обусловлено самой CMS (ни активации пыльников, ни развития функциональной пыльцы), а не аллельным различием между XJH58 и BE4207. Это подтверждается тем фактом, что rf4-bHLH (аллель-невосстановитель), по-видимому, экспрессируется на значительном уровне в B73, поскольку существует 25 независимых клонов EST (например: номера доступа BT043393, BT064392 и т.д.), в частности, из этого инбредного растения в доступных базах данных EST. Как упоминалось ранее, инбредное растение B73 не восстанавливает CMS-C, и имеет идентичный аллель rf4-bHLH с инбредным растением CMS BE4207, который, безусловно, также не имеет функционального Rf4.
Пример 5: Охарактеризация гена Rf4-bHLH в других однодольных растениях
Ортологи Rf4-bHLH однодольных растений были получены из сорго (Sb03g011940), брахиподии (BRADI2G11260) и риса (Os01g11870), все из которых доступны в National Center for Biotechnology Information (NCBI). Эти ортологи сравнивали с последовательностями кукурузы. Как показано на ФИГ.11, остаток Tyr (Y186) в домене bHLH является консервативным среди всех видов, причем примечательным исключением является замена Phe (F187) в аллеле-восстановителе кукурузы. Другие три изменения расположены в различных вариабельных областях и, как правило, не являются настолько же консервативными при сравнении среди видов. ФИГ.11. Эти результаты указывают на то, что F187 в аллеле-восстановителе может играть ключевую роль в восстановлении фертильности цитоплазмы CMS-C.
Белковая последовательность bHLH кукурузы в высокой степени сходна с ортологами однодольных растений из Sorghum bicolor (Sb03g011940; 84% идентичность), Oryza sativa (Os01g18870; 64%), и Brachypodium distachyon (BRADI2G11260, 63%), и в меньшей степени с белками с доменом bHLH из двудольных видов (например, At2g31210 из Arabidopsis thaliana; 24%). Согласно Pires and Dolan (2010), выше, семейство белков растений bHLH можно подразделить на 26 подсемейств, исходя из гомологии последовательностей, идентифицированной среди 544 белков bHLH из девяти видов наземных растений (не включая кукурузу) и водорослей. Если бы он был включен в анализ, Rf4-bHLH из кукурузы был бы отнесен к подсемейству II, которое состоит из белка риса, OS01g18870 (описанного выше) и 10 других белков. Аналогично, Carretero-Paulet et al. (2010), выше, идентифицировали 32 подсемейства bHLH, и Rf4-bHLH кукурузы может быть отнесен к их подсемейству 9, которое также состоит из OS01g18870 и 10 других белков. Большинство представителей в подсемействе II Pires и Dolan, и подсемействе 9 Carretero-Paulet имеют одни и те же последовательности.
Rf4-bHLH представляет собой фактор транскрипции и его предполагаемым местом назначения является ядро. В действительности, несколько предсказывающих программ, включая PredictNLS™, идентифицировали два устойчивых сигнала ядерной локализации (NLS) в Rf4-bHLH: 153-GRKRGRA-159 (SEQ ID NO: 229) и 229-KKRRRR-234 (SEQ ID NO: 230) (в соответствии с положением аминокислот Rf4-bHLH XJH58). ФИГ.9.
Все гены Rf, клонированные к настоящему времени, кодируют белки, нацеленные на митохондрии (Liu et al. (2001), выше; Akagi et al. (2004), выше; Fujii and Toriyama (2009), выше), и предположительно взаимодействуют непосредственно с транскриптами CMS. Однако ожидаемая ядерная локализация Rf4-bHLH делает маловероятным, что Rf4-bHLH нацелен на митохондрии и имеет прямое взаимодействие с транскриптами CMS-C. Вероятно, Rf4-bHLH функционирует выше, активируя и повышая экспрессию ядерного гена, который кодирует нацеленный на митохондрии белок, который в свою очередь приводит к препятствованию экспрессии транскрипта CMS-C, восстанавливая мужскую фертильность. Этот предполагаемый митохондриальный белок может быть представителем семейства белков PPR, или чем-то сходным с Rf2 кукурузы, RF17 риса, или является новым белком. Для идентификации прямых мишеней Rf4-bHLH можно использовать иммунопреципитацию хроматина, установление генной сети и другие молекулярные подходы.
Пример 6: Введение гена Rf4-bHLH в кукурузу CMS путем трансформации
Ген-восстановитель Rf4-bHLH (например, SEQ ID NO: 220) трансформируют в линию CMS кукурузы. Трансформированное растение кукурузы экспрессирует полипептид Rf4-bHLH SEQ ID NO: 225, и Rf4-bHLH является достаточным для восстановления мужской фертильности в цитоплазме CMS-C.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Dow Agrosciences
Ren, Ruihua
Nagel, Bruce A
Kumpatla, Siva P
Zheng, Peizhong
Cutter, Gary L
Greene, Thomas W
Thompson, Steven A
<120> ГЕН-ВОССТАНОВИТЕЛЬ RF4 ДЛЯ ЦИТОПЛАЗМАТИЧЕСКОЙ МУЖСКОЙ СТЕРИЛЬНОСТИ
(CMS) C-ТИПА КУКУРУЗЫ, МОЛЕКУЛЯРНЫЕ МАРКЕРЫ И ИХ ПРИМЕНЕНИЕ
<130> 2971-P10065US (69742)
<160> 230
<170> PatentIn version 3.4
<210> 1
<211> 201
<212> ДНК
<213> Zea mays
<400> 1
acctccagag ccccaacgag tacatccgcg gcgtcactct gcggttcctc tgcaggctct 60
ccgagcccga ggtgctcgag ccgctcgtgc cgtccgtgct ygcgaacctg gagcacaggc 120
accaattcat ccggagacac gctgtctccg cggtttccgc gatctaccgc ctcccgcatg 180
gtgaccagct catccccgac g 201
<210> 2
<211> 201
<212> ДНК
<213> Zea mays
<400> 2
gcgtcgcgag cgtgcgtaca tgaagatgtc gtctgttgcc tgctgctgct gctgcggcgg 60
cgcgggccgg ccctcctccg tccgctcgtc tgaactcccg ygcgtctccg cgcgcaccac 120
gccagcgccg tggtcgtcgt cagctgagcc gaagctccag agcgcccagc cgccgctctg 180
gtcgtccaac agctcgccgc c 201
<210> 3
<211> 201
<212> ДНК
<213> Zea mays
<400> 3
cccgaactcg aacgtctctt tctctcctca aatcagataa ccgaacttgc tgtttgacgc 60
taacatccag ctgcgccttg atctcctcca gatgagcgcc rccatggcgt ggtatccatt 120
accgcagagc ggaggcacag gcacgccgtc gggcggcggc gagctgttgg acgaccagag 180
cggcggctgg gcgctctgga g 201
<210> 4
<211> 201
<212> ДНК
<213> Zea mays
<400> 4
cagcggtgct ggccatcaag gccctgcaca gcaggtgcct gataccgcgg ccgcccttcc 60
tcgccgcggc gcgcgactcg ctgctgtccg tttgtgctgc rgcgccacgc agcggcagcg 120
gcggcggaga gagcgacctc gcgcgcggcg cgccgccgtc gctgcgcacc ttctccagcg 180
ccgccgcgaa cgggtcgaag c 201
<210> 5
<211> 201
<212> ДНК
<213> Zea mays
<400> 5
tcgccgcggc gcgcgactcg ctgctgtccg tttgtgctgc agcgccacgc agcggcagcg 60
gcggcggaga gagcgacctc gcgcgcggcg cgccgccgtc sctgcgcacc ttctccagcg 120
ccgccgcgaa cgggtcgaag cccctgtgct tcctgcatcc ggcctgcagc gccgcggccg 180
acggcgtggg cgacgtcgcg g 201
<210> 6
<211> 201
<212> ДНК
<213> Zea mays
<400> 6
gaaggagcct gccggcgacc gacctgtaga agccctcgga ggcgccggcg gggacgaggc 60
agtaggcgtt ggtgagcctc gcgaagacgc ccacctcgct wgagttctcg aacttgacgc 120
ctgcattata tcatttgttg gaccattatt gttggcgttt tgctatggct atcaatcaat 180
ccgcgcgctg tttgtaacca a 201
<210> 7
<211> 201
<212> ДНК
<213> Zea mays
<400> 7
cactgaggtc tgagaggggg taccgtacct ggaacacgac gtcctcgagc tcccggagca 60
ccagcgcttg gaagaggaag cccccttcat ccgcgacgga ygacctgctc cttttcgtcg 120
cagcaaagct cgccccatcc tggcaccaac ggaacgaaca cccagggctc ggctcatcag 180
agcagtgccg tgcgtgatcg a 201
<210> 8
<211> 201
<212> ДНК
<213> Zea mays
<400> 8
agcaaagctc gccccatcct ggcaccaacg gaacgaacac ccagggctcg gctcatcaga 60
gcagtgccgt gcgtgatcga gtcgagagca gcaccacacg ycacttgcgc aaaccagtac 120
ctggtcctca ggcgtgagcg tgagatccgg cgccgcgctc tgcagctgca ggtcgtggct 180
gaagagaggg aagccgcagg t 201
<210> 9
<211> 201
<212> ДНК
<213> Zea mays
<400> 9
cccgttcact ctctcctcct cccctccgct ccctcccagt cccagatccc gattcgccgc 60
cgcctccggc agatccagcc agcagctccc gccgcagaac ygccgcgggc gccatggaga 120
agccatgctc tctcctggtc cacttcgaca agggctcggc atcgatggcg aacgagatca 180
aggcggatct ggagggcagc g 201
<210> 10
<211> 121
<212> ДНК
<213> Zea mays
<400> 10
ttgcattctg ctaaagaata cggcgggata ataatgtgtc ctagtcttgt aatgaactac 60
wagtgtcttt gcatttggac tggttcgctt gtgagtcata gagattgctg aactcgtgag 120
t 121
<210> 11
<211> 201
<212> ДНК
<213> Zea mays
<400> 11
cgcatcacac ctccgatcag ctcccccttt gccagcatgt ggatccgacc cggacagcca 60
aacccatacg acgtgcacca ccgcggccgt tctcaccaag ycgccatcga cttgctgacg 120
acgacgacgt tcgtcaggcg gtgcgcgctc agaccaccat ggccagcgtc ggcgacgacg 180
cgccgctgct cggcggcggc g 201
<210> 12
<211> 201
<212> ДНК
<213> Zea mays
<400> 12
ggcggcagcg gagagagctc gagggcgtgg agcagacggg gagaccagga aggaaacggt 60
cggggagagg aggagcgcca tgggtgggtg tggcattctt kgccttgctg cggctgcgcc 120
tgcgagaggt agggataata ataaccggat aagccttgga gagcagagaa tgctttccgt 180
ccgatcggca cttaacggtc a 201
<210> 13
<211> 201
<212> ДНК
<213> Zea mays
<400> 13
accaatgatc gtttacccgg catttgcaca tctgcagagc tgcatttcgt tgcttgttct 60
ttcttgattt tgttgatctt gtgtagccag agcaatctat mcatcctttc ctgatctcct 120
ggagtgagtg cagggcaggc aggcaggcag gcgatgcgac gaagaataag tatctgctga 180
ctgttgcaat agcgctactt g 201
<210> 14
<211> 201
<212> ДНК
<213> Zea mays
<400> 14
ctttcttgat tttgttgatc ttgtgtagcc agagcaatct atccatcctt tcctgatctc 60
ctggagtgag tgcagggcag gcaggcaggc aggcgatgcg rcgaagaata agtatctgct 120
gactgttgca atagcgctac ttgtagtagt cacactgatg gaggctccag gaatgttact 180
agtagcagcc ttccaggttg c 201
<210> 15
<211> 201
<212> ДНК
<213> Zea mays
<400> 15
tctccttcac cgacagcctc gtcggccgcg acggcaagct ctaccacggc gccgccacct 60
tctggggctt ctacccgttt aactacacgg ggacccgcga rcagcgggag gcggcgttcg 120
atgacgaaga cctctcccgg ttccgggtca cggcgctgga cttcgtgcac gccttcttct 180
cggcgctcgt gttcctcgcc g 201
<210> 16
<211> 201
<212> ДНК
<213> Zea mays
<400> 16
ctgtcgtttg ttgtcccata ctcccatctg ttctccgcga ggcgcaacga aggcacaaaa 60
cccttacctc catcaaaccc tagcgctcgt gtccccgttt wgctcccgtc gccgctacca 120
tgggctcagg taatccgatc cctccgactc gtgggctaaa ttctctatta tttgtggctt 180
gttctcttgc ggagaatttt t 201
<210> 17
<211> 121
<212> ДНК
<213> Zea mays
<400> 17
aaattgttct tgcttcccga tttccagtcc tctgtcgttt gttgtcccat actcccatct 60
sttctccgcg aggcgcaacg aaggcacaaa acccttacct ccatcaaacc ctagcgctcg 120
t 121
<210> 18
<211> 201
<212> ДНК
<213> Zea mays
<400> 18
tgcagcccgg ctgcaacaaa tttttttcca gtattccagg caatgcggag cccgccgaag 60
ccgaagagaa cggcagccgg ttgccgccat ccttgcagtg sctcaacagc ctgttctcgc 120
catcggcgca gtcctccggt agcggcagcg gcagttcgca tcattgggag aattgaacaa 180
taatgcaata taagaatgga t 201
<210> 19
<211> 201
<212> ДНК
<213> Zea mays
<400> 19
tgccgctgcc gctaccggag gactgcgccg atggcgagaa caggctgttg agccactgca 60
aggatggcgg caaccggctg ccgttctctt cggcttcggc kggctccgca ttgcctggaa 120
tactggaaaa aaatttgttg cagccgggct gcaaaccagg ccgtttgcag cccggctgca 180
acaaattttt ttccggaata c 201
<210> 20
<211> 201
<212> ДНК
<213> Zea mays
<400> 20
tcctgagtcc tgaccctcct ccgtgcgctg cgcaggcatg aggctcctcc tcgtcggcgc 60
cctactccta tgtgccaccg cggcggtgga gtccaagccg mccctcgaca ccctgggcat 120
acccccgcaa ggttcaaccg gatctgtcca cgcctgctct gctatttctt tccatctttc 180
cctgtttcct tcggtcgatg c 201
<210> 21
<211> 133
<212> ДНК
<213> Zea mays
<400> 21
gtggggtcta tcctcatttt tgtgaggggc tgyaaacggc ctggtttgca gcccggctgc 60
aacaaatttt tttccagtat tccaggcaat gcggagcccg ccgaagccga agagaacggc 120
agccggttgc cgc 133
<210> 22
<211> 153
<212> ДНК
<213> Zea mays
<400> 22
gttgagccac tgcaaggatg gcggcaaccg gctgccgttc tcttcggctt cggcgggctc 60
cgcattgcct ggaatactgg aaaaaaattt gttgcagccg rgctgcaaac caggccgttt 120
gcagcccctc acaaaaatga ggatagaccc cac 153
<210> 23
<211> 201
<212> ДНК
<213> Zea mays
<400> 23
ccggttgccg ccatccttgc agtggctcaa cagcctgttc tcgccatcgg cgcagtcctc 60
cggtagcggc agcggcagtt cgcatcattg ggagaattga rcaataatgc aatataagaa 120
tggatgattg ttgctgcttg agcccattgg tgccaatgta cataggaaga gcaggagcat 180
gccggcatga aaaatttgga g 201
<210> 24
<211> 201
<212> ДНК
<213> Zea mays
<400> 24
ccgatcttgc tacccaccgc tccctgcctt tttatgatca cctcccgttc aatcggaggc 60
gggcctctct cccccgatcg gattccgctc gaatctagag rgtttcctcg gatcaaatcc 120
cgctcgtggg gacgggtggt ttcgtgtata gtttgagttg ttaggcacca tgcaggcgtc 180
tttccgtccc gcgacgatca g 201
<210> 25
<211> 201
<212> ДНК
<213> Zea mays
<400> 25
ttcgagctgc gagatggccg ggtaagcacc gatagaaagc aacctcacgc aagctgctcc 60
tgatccgcct aatgagggca gagggcttac aattgcgcca ygttgcattt catgaatcgg 120
cttgtataat tcctgttgtc tggtttggtt tcctggcgtg cagctggcgg agctgcggcg 180
gccgccctcc gcggacgggt g 201
<210> 26
<211> 201
<212> ДНК
<213> Zea mays
<400> 26
gctccccagg gacggccatg cggccatcgc cgctgtccct ggccggctcc ctgttcatgg 60
tgtccgagca cggggggctg gtggagtacc acttcagccc rcaggacggg tgggagtggg 120
tagagcacgg gacgccccac cggggcgtga ccctcgtcgg cgcccctggc ccgtgcttcg 180
acggctccca gctgttcgtg g 201
<210> 27
<211> 201
<212> ДНК
<213> Zea mays
<400> 27
ggccatcgcc gctgtccctg gccggctccc tgttcatggt gtccgagcac ggggggctgg 60
tggagtacca cttcagccca caggacgggt gggagtgggt rgagcacggg acgccccacc 120
ggggcgtgac cctcgtcggc gcccctggcc cgtgcttcga cggctcccag ctgttcgtgg 180
tcggctccga cgggcacgtg t 201
<210> 28
<211> 201
<212> ДНК
<213> Zea mays
<400> 28
tccccccact cgcgtttacg tacacgcatt cgcgaccctg tttccgattg gaattgattg 60
ggtcaattct gtactggaat ttgaaaccca ttatgatcga ygttgcgtta cataggagtt 120
ttggcacctg gccagtcgca atgctgggcg tggggaagag caagacactt gtggaagggc 180
cggtttgtga caacagccag g 201
<210> 29
<211> 201
<212> ДНК
<213> Zea mays
<400> 29
tggccagtcg caatgctggg cgtggggaag agcaagacac ttgtggaagg gccggtttgt 60
gacaacagcc aggtagtggg atggcacacg aacgagagga scaagaggca acgaaggttt 120
catgtcaata cttcgggctt tgctttcaat agcagtatgc tctgggacgc cgacaagagg 180
gctcgtcagg cgtggaatta c 201
<210> 30
<211> 201
<212> ДНК
<213> Zea mays
<400> 30
actactagag cccttccccg atcctttttg tttagtagtt tatttgttga tcatagtact 60
agtaaatagt agtacacgga caaagccatt tgattaatgt ktccgtggat ctttgcctac 120
cctgcacgca caaagcacca acagttgcat ttccgatgac tcccggactt ccacacgtac 180
gcgcgaggct caccggccgt g 201
<210> 31
<211> 201
<212> ДНК
<213> Zea mays
<400> 31
gcatcaggac gccgccgagg aggtgtacca cgtggacgat gtcgacgagg gcagcggtga 60
cggccgtggc cacggcggcg ccggccaggg gaagctgtgc rccaggggcc actggcgccc 120
cgccgaggac gccaagctca aggagctcgt cgctcagttt gggccccaga actggaacct 180
catcgccgag aggctggacg g 201
<210> 32
<211> 201
<212> ДНК
<213> Zea mays
<400> 32
agcaagtata ctgtacttcc gttgttgcta tacgatgccg gctggaccgg aagctacatg 60
atataagcaa tgttgtgctg atcattcacc ctccaagcta yaaataaaca atgcaatgat 120
catcactatc ggttcatcac tgtggcttga ctaggtcacc aactttattg tcaacaattt 180
ttttatttaa aatcattttt a 201
<210> 33
<211> 201
<212> ДНК
<213> Zea mays
<400> 33
ccgttgttgc tatacgatgc cggctggacc ggaagctaca tgatataagc aatgttgtgc 60
tgatcattca ccctccaagc tacaaataaa caatgcaatg rtcatcacta tcggttcatc 120
actgtggctt gactaggtca ccaactttat tgtcaacaat ttttttattt aaaatcattt 180
ttatgataaa atatttgatc t 201
<210> 34
<211> 201
<212> ДНК
<213> Zea mays
<400> 34
aagcgcaggg ttggttaaaa cagatttgtt ggacattagc agttctggtg aggtttgact 60
accggagttg ttgggcgtcc gtcgtattaa ccctgacgtg ygcgtgcgtg cgtgtgttga 120
gctaattaaa cgcagatcgg cgccggggtc aggcctgcac tgctgcatcc ttcccagcag 180
cggctggtga aatggttgtg c 201
<210> 35
<211> 201
<212> ДНК
<213> Zea mays
<400> 35
aacagatttg ttggacatta gcagttctgg tgaggtttga ctaccggagt tgttgggcgt 60
ccgtcgtatt aaccctgacg tgtgcgtgcg tgcgtgtgtt sagctaatta aacgcagatc 120
ggcgccgggg tcaggcctgc actgctgcat ccttcccagc agcggctggt gaaatggttg 180
tgcgcaccgc cgagtctggg g 201
<210> 36
<211> 201
<212> ДНК
<213> Zea mays
<400> 36
ctgttcttga atcggccgca gatggtgttc tcttcgtagc cctggtccgg cccggtacag 60
tccagtagcg gctctgtaaa ggagtgatga agctgcgtgc wgtgcagagg tggcagcagc 120
gtgatgtgct gctgctgctt gtcgctttgc ttgcgcgagt cctagcaaca aggggggggc 180
gttgcaacag cagtttcgtt g 201
<210> 37
<211> 201
<212> ДНК
<213> Zea mays
<400> 37
ttcacatcac cccgatccac cagcagcagg aaccactttc ggttgacaga tggttgaagc 60
gagtagctag gtgtgacggc ccaagcaaaa agtctcgttg mgttaagttg ctgcgtgcag 120
ctactactct cctcacacga atgtacgtac gtacacatgc atgcatgtag tatatatgta 180
tgtacacttg tgatatagat a 201
<210> 38
<211> 201
<212> ДНК
<213> Zea mays
<400> 38
caaggagcag gacagggttg cgttgcgttg cgtcggtctc aacgcttttc gcttcgggtc 60
ccgccgccgg acgtctcttt cggagatcat cctatctgtc scatctacgc gtgtcgtcct 120
gaccgaaagg gcaagcaaca cggtggccaa ggggcctcac tcaccgaccg ccgagcgccg 180
agaaaacaac gggacacggc a 201
<210> 39
<211> 201
<212> ДНК
<213> Zea mays
<400> 39
acgacgacga ccggcatcat gagctcacgg tcgtatccat cttggaggcg tcttcgatgc 60
ccttgaaggc cacacggagc ttcctcaacg aggacacttt yttcccgttg ttctctgcca 120
tgcccgtggc atggacagtg gtgtatggga cctctttgag aagctcaaac atggggatgc 180
tcaggtttgg ccgtgtcatc t 201
<210> 40
<211> 201
<212> ДНК
<213> Zea mays
<400> 40
agcttcctca acgaggacac ttttttcccg ttgttctctg ccatgcccgt ggcatggaca 60
gtggtgtatg ggacctcttt gagaagctca aacatgggga ygctcaggtt tggccgtgtc 120
atctgctcca gcacctccgg tgactgcatg tacaggttgt taccttcgtg cgtcacgctg 180
gctttcgaca tccgcaactt g 201
<210> 41
<211> 201
<212> ДНК
<213> Zea mays
<400> 41
acgccaagat caggagcggc gagatccggg tgctccccgc cgccgtcaag agcgtccgcg 60
ggaacgtcgt cgagttcggg gacggcagcc gccacccttt ygacgccatc gtcttcgcca 120
ccggctaccg gagcactgtc aggcggtggc tcaaggtacg ccgccgcgcg ccgcacgctt 180
taactaacta ccgcatcatc t 201
<210> 42
<211> 201
<212> ДНК
<213> Zea mays
<400> 42
ctacccgtcc gattgccatc cctagtcctg tcacaaaaaa atgctgcctt tgctgttact 60
agtcctgctt aaattaggcc gacccgacca taactgcggt ytgcgcggct tgctggtaaa 120
tgcgagcgga ttagttggac gagcgaccgt gaaggcccca gccggcggcc gtacctgtac 180
tacgtcctcg gtcctctccc t 201
<210> 43
<211> 121
<212> ДНК
<213> Zea mays
<400> 43
aagtggcttt tgtctgatgg gcggcggtgc atcgtctgtt tcattcatca gatcgcacat 60
rtacatttta ctacagtgca gtgcaggtga ctgtcctgtg gcaacgggta cggttatgag 120
t 121
<210> 44
<211> 201
<212> ДНК
<213> Zea mays
<400> 44
catcctgcat accccaccgt agtggcagcg agccagcgac ctcgagtagc agcagaggct 60
ccgcaggggc gaaaaggtgt gaagcttcct tcggagatcc magtagcaga agcaacgcag 120
gcatcggctt gcgacaggag aggacggaga tgacgcagaa gcagaccctg ttcaaggggc 180
agagcaagaa gaagactatc c 201
<210> 45
<211> 201
<212> ДНК
<213> Zea mays
<400> 45
attgagttcc gcctcccgct tccgctccag cccctcgtcc agtagcttcc gcttctacgg 60
cacggcaata gattaagcgg aaatcaataa ttgcagtgtc racccaccgc gcgcggccat 120
agaaacggga tggggaagaa gcggacgtgc ccgcagtacc ttcagagcag tgtacttctc 180
gtggaacttg tagtcgagct g 201
<210> 46
<211> 121
<212> ДНК
<213> Zea mays
<400> 46
tgacgtggat ggttttgttg ggttgggttc ccagtctggt gctgtagcgg tgtagcagga 60
wcgcgtgggt agccttgagc ctggcgattc tgtaattgtc ctttgagttc tgaaatgtta 120
a 121
<210> 47
<211> 121
<212> ДНК
<213> Zea mays
<400> 47
taagagattc atttttacat gactttttgt tttttcattt ttacactctg gatatggacg 60
rgactagttg aatttaggaa gatcgtatga tctcgaaaat gttcagtaac tgcatcaaca 120
c 121
<210> 48
<211> 201
<212> ДНК
<213> Zea mays
<400> 48
ttttcgcaaa taatggtcgc cggaatcttg acctgtgccg aactgccgat gccgtcgcag 60
cgatggcagg acagatacac catcaatgct aatgctcgaa mcaacaggac acatgcgcat 120
ggctgcctgc cggtgagccg acggtccagc ttgtgttcag atctccccca agggaccggc 180
gctacctagt taagagcaag t 201
<210> 49
<211> 201
<212> ДНК
<213> Zea mays
<400> 49
gcacaggaat tcaaacgaat cacggagcac ccggagggag accgatcgag aagcaacgga 60
aggccaggga gaggccgctg acctcgcgcc tgttgtgctc rtcgacggcg aagcggccga 120
gcccgtcgga ctcggcgctg ttggccgcgg ctgggttctc cttcacgccg ccgaggacgt 180
ggccggccat ggctgctcct c 201
<210> 50
<211> 201
<212> ДНК
<213> Zea mays
<400> 50
agcacatacc ctcttactac tcttgaggat ttgcttcctc catccaaaag aaataattgc 60
acatgtcaaa caatccgcac taaggcattt ctacaaacac rctactgcac aggccaccca 120
ggctccagcc cttgtactcg ctatgcagac gcatcaacag tggtagcaat gtctccatct 180
cataagcggg agcacgaacc t 201
<210> 51
<211> 201
<212> ДНК
<213> Zea mays
<400> 51
tgggacgtga agccgcagga aactacgggg cgtccttctc gggtagtggt cccgctggac 60
cacggacggg acggcggccg tcgtcgtcgc gtaatccgcc ygtattatat gcccgtgtgt 120
cggaaggaaa catggcctgt tcttcttccc agcgcagacc taatcatgct gccattgccc 180
cgggtggcgc atagcttcaa a 201
<210> 52
<211> 201
<212> ДНК
<213> Zea mays
<400> 52
ggcctcgacg gcctcgagcg tgaggtggtg cagcgtgccg gccaccacct gctccttggc 60
ctccaccacg cgcacgaact ccagcagcgc gttctgaagc maggcgcaca gatgtgcatc 120
gacagaaccg tcccccccag atcagcacag gaattcaaac gaatcacgga gcacccggag 180
ggagaccgat cgagaagcaa c 201
<210> 53
<211> 201
<212> ДНК
<213> Zea mays
<400> 53
gcgtggccag ctgctccagc gtcaccccgc gcctgtacca ggggaacgcc accagcgtcg 60
acccgctgct ggactccgcc tacgcccgcg cgctcatgtc stcgtgcccc aacccgtcgc 120
ctgcgtccgc caccgtgggc ctggacggcg gctcgccggc caggttcgac agcgggttct 180
acgcccgggt gcgccagggc c 201
<210> 54
<211> 201
<212> ДНК
<213> Zea mays
<400> 54
cggggcgtcg ctgtggcagg tggagacggg gcggcgggac ggcggggcgc cgtcgctggc 60
gtccgacgcg ttgggggcgc tgccgtcgcc cttcgcgggg ytcggcggcc tgctggcggg 120
cttcgccagt cgcgggctca acctgacgga cctggtggcg ctgtcggggg cgcacaccat 180
cggcgtggcc agctgctcca g 201
<210> 55
<211> 201
<212> ДНК
<213> Zea mays
<400> 55
ggcggcggga cggcggggcg ccgtcgctgg cgtccgacgc gttgggggcg ctgccgtcgc 60
ccttcgcggg gttcggcggc ctgctggcgg gcttcgccag kcgcgggctc aacctgacgg 120
acctggtggc gctgtcgggg gcgcacacca tcggcgtggc cagctgctcc agcgtcaccc 180
cgcgcctgta ccaggggaac g 201
<210> 56
<211> 201
<212> ДНК
<213> Zea mays
<400> 56
ctagctagat ggacataaat aaaaatctac tcactactag ctccgaggcc gtatttattt 60
gaatagcctc agtctattta ttaccatgcg ctaattttac ygcaattatc tgcagtaggg 120
gatcatggta aattgtcaga gtttggaggc gttcgcggta actttgctca gagcggtcgc 180
tgccttggac ccagactggc g 201
<210> 57
<211> 201
<212> ДНК
<213> Zea mays
<400> 57
tttgttggga aatctaggga tcaatagagt accgcaggtc cgcagcaaca acgactcaac 60
gagtgggcag ttcaccgtgc tggcggcaca atacctcccg ycgtcggggc actctgccgc 120
caaatcgcga acccgtgcca ttagcgaaca cgtattaagc atggctgcga tcaaacagta 180
aaagttgata tagattagag t 201
<210> 58
<211> 201
<212> ДНК
<213> Zea mays
<400> 58
gagtaccgca ggtccgcagc aacaacgact caacgagtgg gcagttcacc gtgctggcgg 60
cacaatacct cccgccgtcg gggcactctg ccgccaaatc rcgaacccgt gccattagcg 120
aacacgtatt aagcatggct gcgatcaaac agtaaaagtt gatatagatt agagtcaaac 180
gaacactagt atgtctttct t 201
<210> 59
<211> 201
<212> ДНК
<213> Zea mays
<400> 59
tgggcagttc accgtgctgg cggcacaata cctcccgccg tcggggcact ctgccgccaa 60
atcgcgaacc cgtgccatta gcgaacacgt attaagcatg rctgcgatca aacagtaaaa 120
gttgatatag attagagtca aacgaacact agtatgtctt tctttagcaa aagctttctt 180
tagaatgtaa aggagtgttt g 201
<210> 60
<211> 201
<212> ДНК
<213> Zea mays
<400> 60
cgtggtcgtc gccgtcgccg ggggcgactt ggcagggtca gcgtgcgagc aggtccattg 60
gtagtcgccg atcttgacgc tgaacctaac ctgcgtgtcg stcttctcct cgagaaggat 120
ccagtaccgc gccgagagca cgggcctcgc cgccgccgcc gcgctccggg cgcggcgacc 180
tgccgcgcgc aagcgcaacc c 201
<210> 61
<211> 201
<212> ДНК
<213> Zea mays
<400> 61
acggagtcaa ccggacggcc gccggttctt cccaggtgga tcacgtccgg cgccgtcgtc 60
ggcatgcagg gtggcacaga cgccgtccgc agagtttggg rccagctcaa ggactacgac 120
gtccctgtct ctgcgttctg gctacaggtc tgaggaggaa ctgacagttc ttcgtgtctg 180
acgacgcacc ctgaaccggc c 201
<210> 62
<211> 201
<212> ДНК
<213> Zea mays
<400> 62
atatgtccca cccgtccgac ggcgagctca cctacatgcg cgcgcatttc gaccgcctcg 60
tcggttccaa ggactccgag acgtactaca tgcacaaccc mgagggcggc accgccgggc 120
cggagctcac aatcttcttc attaggactt agaatcaaat taaaccaaca acctcatctc 180
tggtatgtgt tctttcctgc c 201
<210> 63
<211> 201
<212> ДНК
<213> Zea mays
<400> 63
tctccgtccc gacaaggaac tgctggtacg tgagcgcctg cacgcgctcg tccgccgggt 60
agtgcaggaa caggtggcgc gccacgggga gccccgtccg mgcggcctcc ccgacgaggc 120
gcgcgcggta gaactcccag gccttgtaca tcttggcgca gcgcgcgaag tgcgccagcg 180
tgcggccgct ggagtagaac t 201
<210> 64
<211> 201
<212> ДНК
<213> Zea mays
<400> 64
tctggctaca ggtctgagga ggaactgaca gttcttcgtg tctgacgacg caccctgaac 60
cggccctgaa ctgcttgctt gctgcacgtt ggctgggtct ytggctgcag gattgggttg 120
gccagaggaa gacggcgatc gggtctcagc tctggtggaa ctgggagctc gatgatgccc 180
actacaatgg gtggaacgat c 201
<210> 65
<211> 201
<212> ДНК
<213> Zea mays
<400> 65
ctctccgccg cggcacggcg gcggctggct ctccactcgt cggcgatctc gcggttgacg 60
cgcgcccaca gctccgggta ccggttgtgc gcggcgacgg rgtcctcgcc cgagtgcagc 120
cgcgcgtcca gcggcaggcc ctcgccgaag tcggccatcc agccgctgac gccgcggtcc 180
accatccccc gcaggacggt g 201
<210> 66
<211> 201
<212> ДНК
<213> Zea mays
<400> 66
ctcgtccatg gccggccgca gctccgcgct gtccgacacg caccgcgccg ccacggcgag 60
gacgcggtcc gcgcacgccg tgggcggcgc ggccaccctg scgtccagca cctcccgcgc 120
gcgcccggcg ccgatcagcg gcgcggccca ggacacgatg gacgacgggc acgagtccac 180
gtccatcacc ctgcgcccgc t 201
<210> 67
<211> 201
<212> ДНК
<213> Zea mays
<400> 67
gcgcgatgca tgccacgggg actctgtacg gccaccgccg ggcgcgcatc accatcgcgc 60
tgcacgagag ccccggtagc ccgccgtgcg tgctgctgga ygtcggcgtg cccacggcca 120
agttcatcca ggacgtgagc gcggcgggga tggtgcgcgt cacgctcgag tgcgagaagc 180
agcaccatac ggtggacgtg g 201
<210> 68
<211> 201
<212> ДНК
<213> Zea mays
<400> 68
actctgtacg gccaccgccg ggcgcgcatc accatcgcgc tgcacgagag ccccggtagc 60
ccgccgtgcg tgctgctgga tgtcggcgtg cccacggcca rgttcatcca ggacgtgagc 120
gcggcgggga tggtgcgcgt cacgctcgag tgcgagaagc agcaccatac ggtggacgtg 180
ggggcgcgga ggctgctcga t 201
<210> 69
<211> 201
<212> ДНК
<213> Zea mays
<400> 69
gccagcgtgg gttcattttt gctacctgag ctacccgtgc ccagattatt gtccagctcc 60
agccgcctaa caaccacgta cgtacgcgtc tcttgcacga sgtgaacaac gatcaaatta 120
aacagccgta gattagtcag tcagtcagtc tcctaactgc gcgctgtgac agtaacagta 180
gcaagcaaca gtaaacatcc g 201
<210> 70
<211> 201
<212> ДНК
<213> Zea mays
<400> 70
acgcgtctct tgcacgaggt gaacaacgat caaattaaac agccgtagat tagtcagtca 60
gtcagtctcc taactgcgcg ctgtgacagt aacagtagca mgcaacagta aacatccgtc 120
caaaggtttt accaatcaac tacctgaaaa cgagatccaa ttcgctctcg aatcgagcca 180
acgcgtcctc ttctgctttc g 201
<210> 71
<211> 201
<212> ДНК
<213> Zea mays
<400> 71
tctctccctc caggaacagc ggcctgctca acatgctcag cctcatgaag cgcaaggcac 60
ttgagatcgc cggtggtgac tcaacgtcaa gtcagcaatc wgtccaagag gttaccagac 120
cgcgtctagt gaacgggaaa gagcctgaat ttgcagcttt cgaggtgcaa gaaaaggaaa 180
aggaaaggcc acaggatgca g 201
<210> 72
<211> 201
<212> ДНК
<213> Zea mays
<400> 72
cacacctcca caagggccat gctggcgtgg ctgggaccaa tggggagaca catttcaatg 60
tgagggtggt gtccaaggag tttgagggga agagcttgct yaagaggcac agggctgtct 120
atgatctcct gcaggaggag ctcaagaccg ggttgcacgc cctgtcaatc gatgcaaaga 180
ctccatctga agtttaggta c 201
<210> 73
<211> 201
<212> ДНК
<213> Zea mays
<400> 73
agtccgtccc ggacgcgcgc acccggtaca agcagctcct tgcctatgcc gcgcgcctgc 60
cgcccatgga cccggcgctc aagaccgacg ccaaccgcgt scggggttgc gtctcacagg 120
tctgggtcca cgccgagccc gacgagggcg acgccggccg ccgcagcgtc aggttccacg 180
ccgactccga cgcgcagctc a 201
<210> 74
<211> 201
<212> ДНК
<213> Zea mays
<400> 74
agggcaatca aatcaaatca aacaaagaga tgagttgcaa attatagatg taaaagatca 60
taaacgaagg aaccttaaca gataatttga tcccacctat ktacagacag aataaatgct 120
atagtattac agaagataat aggaacaatt ggatctatac cattaaaaga tccaaacttt 180
agatatatac catggacccc a 201
<210> 75
<211> 201
<212> ДНК
<213> Zea mays
<400> 75
gcaagcaagc aaattttggg tcctagcttg aaggactgcc atcttttttt ttggttggtg 60
agctacacat caaatgttaa tcccccgttt ccgtactctt rtattgtgct tattattatt 120
ctcttgggaa atttggatcc ataccattaa aagatcacca ctttggatcc ataccattac 180
tatctcactt acatgtgggt c 201
<210> 76
<211> 201
<212> ДНК
<213> Zea mays
<400> 76
cgacggatcg aacggccgag cccacgcccc cttgaagtag atggcgttgg cgaggacgag 60
cgccgtggac gagtcgacgg agccgggagg caggacgtcg ycgatgagcc ctttcgttgc 120
tcctttcacg aagtcgttga cttgctgcct tgccgcctcg gagttcttca agaagtcgac 180
ggactccgcg accgccgcgt a 201
<210> 77
<211> 201
<212> ДНК
<213> Zea mays
<400> 77
ccgtccggca acgtgtcgtc cgaaaagcag cacttgacgt cctgcgcaac agcagcagcc 60
ccgcaaccaa gtcatccacc acccgatcaa tccgccgacg rccgacgtgc ggtgcaagac 120
caaccaacta gtgtgtgttc caggaaggtg ccgagttccg aatagctcac gatgggtacg 180
gaagggtaga gccggagggt c 201
<210> 78
<211> 201
<212> ДНК
<213> Zea mays
<400> 78
ccccctacag gccgcatgca aggagaggga gctgcgtagg ggcagccgtt gcagttagtc 60
tcacaacgag ggtccgcacc agcgccagca ccgacaccac racggtgacg agccatcgcc 120
agactccgga cagtgggggt tcagcatctc ctccatccta gtcttcgtcg cgggctcgag 180
gttgtacgag aataaggtgc c 201
<210> 79
<211> 201
<212> ДНК
<213> Zea mays
<400> 79
ggagctgcgt aggggcagcc gttgcagtta gtctcacaac gagggtccgc accagcgcca 60
gcaccgacac cacaacggtg acgagccatc gccagactcc rgacagtggg ggttcagcat 120
ctcctccatc ctagtcttcg tcgcgggctc gaggttgtac gagaataagg tgccctgctt 180
ttcctcaccc gtgggcggcg g 201
<210> 80
<211> 201
<212> ДНК
<213> Zea mays
<400> 80
caccgacacc acaacggtga cgagccatcg ccagactccg gacagtgggg gttcagcatc 60
tcctccatcc tagtcttcgt cgcgggctcg aggttgtacg rgaataaggt gccctgcttt 120
tcctcacccg tgggcggcgg cacgatgtga gcgaacgcgg ccggtcggag gtcagcgcgg 180
cgagcgtgaa cggggtcgtt c 201
<210> 81
<211> 201
<212> ДНК
<213> Zea mays
<400> 81
gggcaggatg cggttcctgt ggggcgcgga cgccgaggag ttcaggccgg agaggtggct 60
ggacgacgcc ggcgtcttcg tccccgagag ccccttcaag ytcacggctt tccaggtgaa 120
atctcacgtt cgaccctgtt ctgtgctgtt gctgtccact cgttttctca gtttacttga 180
tgagctcttg tttgactgtt g 201
<210> 82
<211> 201
<212> ДНК
<213> Zea mays
<400> 82
tcccggcgac gacacgacct tatcatagag atcattgagg gttagggtct caccgtcggg 60
gaggagtaga agcatgtaga acgcgtcctg ctgcgcgccg ycgtcgttct tgtagggcag 120
cttgagcgcc ttgaatccgg ggaacacggc aacccgctgc cgctggtcct cgaaaccgct 180
cgtagtcatg aacggcacgc g 201
<210> 83
<211> 201
<212> ДНК
<213> Zea mays
<400> 83
gttgcgggtt tgcccggatg gatcaggtca tcaggttgag acacgaagaa gcgaagtttg 60
agccgctgtt ccatttccca tactggcggc agacgcgccg magagcctga cgtgcgagcc 120
tgtgccacac tgccccacgg cccccacgcc cggaagtggc tcgatgccaa gaggttttta 180
cgcggcgcgc ggcgggtcat c 201
<210> 84
<211> 201
<212> ДНК
<213> Zea mays
<400> 84
atggaacaga atgctttaat tcaacctttc aaagaaggga agcataaatt attgaccgac 60
ctgctgaact gagctaaaca acaaattgga tcaaaacaaa watacggacg ggcacgaatc 120
tctcacgtgt tcccggaaca aaggacaaag attaggtctc cacagtcggc gtgaagacac 180
aacgaaagct aacgtggtcc t 201
<210> 85
<211> 201
<212> ДНК
<213> Zea mays
<400> 85
atgcctggtc cctggcaggg gagatgcgat gcgaggagct ggtgccagag cgggcttttc 60
cttctccgag cccgagggcg tatcacgtac gggagtggga sacggaggag cggggaagaa 120
ggcgacggga aggaaggcag gcgggtgtgg gaatgtggga agggaactgg gaagtgcgga 180
atcgaagccg agacagggag g 201
<210> 86
<211> 201
<212> ДНК
<213> Zea mays
<400> 86
ggaggaaggc ggtctcggag atgttgaact cggcggcgac ggactgcatc caccgctcgt 60
cggcggcctt ggcggcgtcc tcgaggaggc acaccgcggc kgggttgccc ttgaacggct 120
ccgccgtgaa ggcgtccacc tgcgacgacc acggcgacga tgtcaggtat ccagagagga 180
ggaagaatcg ctcccgctcc t 201
<210> 87
<211> 201
<212> ДНК
<213> Zea mays
<400> 87
cgcggctggg ttgcccttga acggctccgc cgtgaaggcg tccacctgcg acgaccacgg 60
cgacgatgtc aggtatccag agaggaggaa gaatcgctcc mgctcctcca agggagtaca 120
gagcagtatc tgaccactgc gtactggatg ccgtccttgc ccattcctgc gacgaagaaa 180
cagctccggg ctcctgggct a 201
<210> 88
<211> 120
<212> ДНК
<213> Zea mays
<400> 88
cccggacwtt gatcaatgaa tgttttgaag caagaactra ctgagtgtta aaggagctta 60
katcaattta aggagagcgg ttgtgtcaag gctactctta ggtccacagt tgggtgtttg 120
<210> 89
<211> 201
<212> ДНК
<213> Zea mays
<400> 89
gccgcgtccg gagctccggc gggttcacgt tcccggagct cagcctcagc gccggcatct 60
tggacgagcc accgcgcgtg tcgttggagg tgttccgccc satcgacgag gactcggtga 120
tgcccgcgga ccctccgctc gccgccgccg ccgtgccccg tgtcgggcta cccccgcgcg 180
ctccgccggc ggcgctggtg g 201
<210> 90
<211> 201
<212> ДНК
<213> Zea mays
<400> 90
gtgagcggcg agatgttgcc catactgacc atggactgcg caaagtggcg gaagaagaga 60
ccggcgtcgg cggcatacgc cttgacgagg gacgccgtct sggcgctctt ggtcaacaga 120
acctcgtcgg agctgagaag gcccctgccg gccaggaggt tcttgtagta gaagttgtcg 180
aacttggctg gggcgacgac g 201
<210> 91
<211> 201
<212> ДНК
<213> Zea mays
<400> 91
aatatctacg tccaaacgtg atatataagt cgggtccatt aacatcgagt ccggagttga 60
tatgttcgaa atatggacag cccaatagga aagtggcctg satttctgaa tcgacgggcc 120
aggacgatgg atttaaagcg gtccgttttt ccagcccagt agatctcgca gtcatgtatc 180
ggacggctgc ggcctcaatt g 201
<210> 92
<211> 201
<212> ДНК
<213> Zea mays
<400> 92
ggtccgtttt tccagcccag tagatctcgc agtcatgtat cggacggctg cggcctcaat 60
tgctgatcgg atcgcggccg ctggaaatct gcgtgactgc rtctatactg cgcaggcacg 120
agcgtttgtg ttgcggcgag gttttcgtgc ggccgctgcg gtgacaagca ggtcctccct 180
caagctcgcg gcggatcagc g 201
<210> 93
<211> 201
<212> ДНК
<213> Zea mays
<400> 93
cacgagcgtt tgtgttgcgg cgaggttttc gtgcggccgc tgcggtgaca agcaggtcct 60
ccctcaagct cgcggcggat cagcgaccga tcgacccgtc wtcctcggtt cctcatccat 120
cccgggcatc cggattccgg tgagcggagg aaggcgccat gaaagctgcg gaggacgcca 180
cgcgctgccc tcagatcccc c 201
<210> 94
<211> 201
<212> ДНК
<213> Zea mays
<400> 94
agggtgaatg gcgtcgcgct ctcatctact agggcattat ctcagggcag tatactttcc 60
gaactagcaa cgggcacgtg gcctggcact gtagcggccc racatgagaa cggtccagtc 120
cgaatggaag tgtgtttggg ccggcccgac atgtcacatt tggaattaag gataaattca 180
aatgtatctt aaatacgtgt t 201
<210> 95
<211> 201
<212> ДНК
<213> Zea mays
<400> 95
ctcatctact agggcattat ctcagggcag tatactttcc gaactagcaa cgggcacgtg 60
gcctggcact gtagcggccc aacatgagaa cggtccagtc ygaatggaag tgtgtttggg 120
ccggcccgac atgtcacatt tggaattaag gataaattca aatgtatctt aaatacgtgt 180
tagaatcaaa tctcacacat a 201
<210> 96
<211> 64
<212> ДНК
<213> Zea mays
<400> 96
agtagaacaa tggtaaaaga cgacgaaggt gtactcatcg ycccgtgttt tagattatct 60
gcag 64
<210> 97
<211> 121
<212> ДНК
<213> Zea mays
<220>
<221> misc_feature
<222> (27)..(30)
<223> n представляет собой a, c, g или t
<220>
<221> misc_feature
<222> (70)..(70)
<223> n представляет собой a, c, g или t
<400> 97
gacaagaagc tcytcaccgy tgtaagnnnn cgggtacaac agttcctcta cataactccg 60
yctgccttgn agaaactacc ctattattta actatgcaag acycrtccca tcctactcta 120
t 121
<210> 98
<211> 121
<212> ДНК
<213> Zea mays
<400> 98
agctgagagc aacatgaacg atctggtgtc tgagtaccag cagtaccagg atgccactgc 60
mgatgaggag gagtacgagg acgaggagga ggtccatgac gagtaaggtg atcttggacc 120
c 121
<210> 99
<211> 77
<212> ДНК
<213> Zea mays
<400> 99
ttaacatcgt ctctctatgg acagatgcag tagaaagwgt gtatctgaag aaaatccggc 60
acatgcggtt ggtgcgt 77
<210> 100
<211> 75
<212> ДНК
<213> Zea mays
<400> 100
ctgcagagca gctgccattg tcccgtgaag aactrgggcc gggcctggag ttgcagttca 60
atcgtgttgt tactc 75
<210> 101
<211> 70
<212> ДНК
<213> Zea mays
<400> 101
aacaagtgaa aactaagctt cctggagcac ccaagtttct tttrtgtgtt ctagcggaaa 60
gaaagaattc 70
<210> 102
<211> 101
<212> ДНК
<213> Zea mays
<400> 102
gttagctgtc cagcttaacc ttccgaatat ttttagatgt aaaggaaatg wgaattgatt 60
gctgcaataa atctaaccag cttaacacag cacattaatt g 101
<210> 103
<211> 101
<212> ДНК
<213> Zea mays
<400> 103
atgcatacgg ttccacatgg accagtacgc gcgtcatgag gagtaaccac rtggatggac 60
cccggtttgg ctgttttaaa taaattagtt aaaaatgttt t 101
<210> 104
<211> 101
<212> ДНК
<213> Zea mays
<400> 104
gggcgtcgtt gaggtgagaa gagatggaaa aaaggcgtga tccaacctcc ragggcaccg 60
gtctaacagc ccgcttgcat tgcattgcat tgcccggtta a 101
<210> 105
<211> 101
<212> ДНК
<213> Zea mays
<400> 105
gataggatcg gatgagtagg ttcacttgct tgagttcacc ggtataattc yggatacatc 60
tggttaggtc atcctttggt cagctgcccg caagcttaac t 101
<210> 106
<211> 101
<212> ДНК
<213> Zea mays
<400> 106
atggagacca aggaaagcat cgcagtccta ttagacagaa acctatttgt ycggggatgg 60
aatgcaagct gctgggcctg ctggacacac tgtctgcagc t 101
<210> 107
<211> 101
<212> ДНК
<213> Zea mays
<400> 107
tgatcaaatg aaactaaatc caatccatgc tccaacatcc gaatctcaga rcatacatct 60
ttgtccgtga caacaaaggg aacagaaagg ctgctttgat c 101
<210> 108
<211> 101
<212> ДНК
<213> Zea mays
<400> 108
agtcgaggct cctgttcttg ttcccaccgg acattctcgc cgcaccggct kggtcaccga 60
tctctttcca caggaaaccg gcttccattt cctcggcctt c 101
<210> 109
<211> 101
<212> ДНК
<213> Zea mays
<400> 109
tcctggggag gcagagccct ccccaccggc atattcacac tcgaaccacc rccgcctctt 60
tttcatctcc cctgtctctt gtctggcgct cggacagcga c 101
<210> 110
<211> 101
<212> ДНК
<213> Zea mays
<400> 110
tccaaggaga accaatcagc catggtctca taaacgctgc agggtctagc yaggaaaggt 60
taagagacaa catctccaaa ttttaactga atccatctgt c 101
<210> 111
<211> 101
<212> ДНК
<213> Zea mays
<400> 111
taaataaggt gttttggtgg aggggtttaa agaatgatgt gacacaatat rtgcagcaat 60
gtgttgtctg ccaaaaggct aagactgaga gagtgcatcc a 101
<210> 112
<211> 101
<212> ДНК
<213> Zea mays
<400> 112
tgggctgtct atcgagcttt attgaaggac tcctgtatcc atgtgctaaa sagaggagca 60
gctgctagat catgctgagc tgataaccaa gagtccactg g 101
<210> 113
<211> 101
<212> ДНК
<213> Zea mays
<400> 113
aagatctggc ggtcacaacc tagccgcgcc gtcgacgagg ttcgttctgc yttgagcttt 60
gttactaggt tttcgcctgt gattccataa ttcgaggaag a 101
<210> 114
<211> 101
<212> ДНК
<213> Zea mays
<400> 114
tgtctggccg cgttacgcaa gaggtggaac ttacggacga aggaagcgac rgcggaggcc 60
atggcgaggt agacgaagtc gagcgagacc atggacaccc g 101
<210> 115
<211> 101
<212> ДНК
<213> Zea mays
<400> 115
ccctgccggc tgcagcaggc acacggtacc cttgtggatg gcactgctgc ratatggcgc 60
gcacgcaaca gctgaggctc ggcacgcgtg cttgcattgg c 101
<210> 116
<211> 101
<212> ДНК
<213> Zea mays
<400> 116
ggccaaagtg gaccgtatat gtacgtgtac tatatcttct cgaactcagc ytgtttccat 60
cttggctctt gcccgccgct gaaaggagga attccaaact t 101
<210> 117
<211> 101
<212> ДНК
<213> Zea mays
<400> 117
tctcttgaca gcagcgagat gttcctggcg tggcgcctcc atgaaccggc wgaggtaccc 60
caccaagtac gccagatccg gcctggagtt gcataggtac c 101
<210> 118
<211> 101
<212> ДНК
<213> Zea mays
<400> 118
gaattttctt gtcccgtgcc ggccgccgtg tctaccctgc gccaccgccc yttccaacgc 60
ctgccgtgaa gacaaaaggt gagcttttta tcccctcttc c 101
<210> 119
<211> 101
<212> ДНК
<213> Zea mays
<400> 119
acgtacgaga tttgctaatg cccgaagtaa tgcaacgaag cagctttgac racgaaggaa 60
gaggacaggc gagcgcgggc gtacgcggcg cacacatccg g 101
<210> 120
<211> 101
<212> ДНК
<213> Zea mays
<400> 120
tttttcctgt tccctttgtt gttcaaatag tttgcagctg ttgctgcact stgtgtgcaa 60
tatgaagctg actttcgacc caacatgagc atcgtggtga a 101
<210> 121
<211> 101
<212> ДНК
<213> Zea mays
<400> 121
aatcgatgaa gggccggcag cggggaattg aagttctgcc ggcagcgggg rattgaagtt 60
ctggtacgtg gtaaaaaaaa acaaagtagt tgtcatggtg t 101
<210> 122
<211> 101
<212> ДНК
<213> Zea mays
<400> 122
atctgtggac gtagatgatg agcggccgga ttatacggcg acgacgcaaa kacagtgaat 60
gcatcatgca tggttggtga gactcaaaaa gactcgcaag a 101
<210> 123
<211> 101
<212> ДНК
<213> Zea mays
<400> 123
tccaatgcca tgcaagaggc ccagcgcacc actggactgg tccggtgccc rccacgtcag 60
atagccattg gagctagtcg ttgctgactg ttgtgtctct t 101
<210> 124
<211> 101
<212> ДНК
<213> Zea mays
<400> 124
actcgaagct gatgacttcc ccggatcact cgatagctac tgatttgaca ycgcgcatgg 60
agatgtcaag gcggggtgac acatcaataa tgggagagcc g 101
<210> 125
<211> 101
<212> ДНК
<213> Zea mays
<400> 125
gtcatcttgc atctaagacg tcggaccggt ttggtgtgca tcggaccggc yatgcagaga 60
ggatgtaaaa tggacacttc gaaggatggg ttctcggacc g 101
<210> 126
<211> 101
<212> ДНК
<213> Zea mays
<400> 126
gaaaaggatg gccggcccta cgacgcaagt tgctgctgct agctgcaccc rtacgtgagc 60
caatgtagta gctttcccat cccctgcatc gatctgctca t 101
<210> 127
<211> 101
<212> ДНК
<213> Zea mays
<400> 127
attcaaagtg ggcatgtgcc ggtccggtcg aataattgtg tcatgcctcg rccgccttct 60
cggcccttag taccggcttg tctcgatacg attatatttt t 101
<210> 128
<211> 101
<212> ДНК
<213> Zea mays
<400> 128
aattgaacca ccatccatcc gagacagcac gcagcaacgc atacgctact rgtcaacgga 60
ggacctcacc gtgatgagga aagatccaca tctcaacaag c 101
<210> 129
<211> 101
<212> ДНК
<213> Zea mays
<400> 129
agacgaagaa tccggcattc agatctaagc ctgcgcaaca tgaagcaaac kgatttgtcg 60
ctaaatcgaa acaaagtagc tcaccgaggg cgcgcgaggt a 101
<210> 130
<211> 101
<212> ДНК
<213> Zea mays
<400> 130
tgctgcgcac cctcgtcacc cccatgtcgt gctcgccgtc atccgtaaga rtgtccggtg 60
tccgggccac gtcgcgaggt aataatgtcc ggtgttccca g 101
<210> 131
<211> 101
<212> ДНК
<213> Zea mays
<400> 131
agatttttcc cggccggtga agcctcgttc gcaacgtaga gcatgcactc scgtcttgcg 60
cactatcatc agctcgagct ccagcctcca gctgctaaaa c 101
<210> 132
<211> 101
<212> ДНК
<213> Zea mays
<400> 132
gcttttggat aaggaaatac tagtggaagt ccagggcatc catgagaggt mgagaaataa 60
gagtacattg ttttacttac ataagccatg gtgtctttta t 101
<210> 133
<211> 101
<212> ДНК
<213> Zea mays
<400> 133
ggtgccgtcg tggtgcgcga actcggcgcg aatatcggct tccgtgatag ktgcgctggt 60
gcgtttttcc ggcggcgcag cgttcctgcc cttggccgcg t 101
<210> 134
<211> 101
<212> ДНК
<213> Zea mays
<400> 134
tcttctcagc accttgtgga gatgtcttct cagtgccttc gcaagcatcg rtcacgtaaa 60
gtgcagctcg tgtttttttg ctgcttggga tctcacgcaa g 101
<210> 135
<211> 101
<212> ДНК
<213> Zea mays
<400> 135
cccagtccca ctccgtccac tcgagctctc caccagcttt ccccttctct mcggtccggg 60
aatcggaagc tcagggcaga aggtaagcta ccagaccatg t 101
<210> 136
<211> 101
<212> ДНК
<213> Zea mays
<400> 136
ttctctctct acctatcggt tcttcttaga tgatcgccgg aatctcaacg ycggtcgtca 60
catattcata ttcatattcg aattaaaaca tggtagatag t 101
<210> 137
<211> 101
<212> ДНК
<213> Zea mays
<400> 137
accattggtg cctcagatcg cgcacgcaca gacacccttg tcttgtgggc yggcaactcc 60
gcccgctcgc cgccgccgtt ctctctcgcc ggcggtgctg t 101
<210> 138
<211> 101
<212> ДНК
<213> Zea mays
<400> 138
gacctggaga agctgctggt acatcgcgcc atagttccgt gtgagctcgt ycatcatgcc 60
acgcagccgc ctgttatcct ccaccacccg cctgagctct g 101
<210> 139
<211> 101
<212> ДНК
<213> Zea mays
<400> 139
agtgtatgta tgtatattta atttgatatg tggttagcat gctcaataga matgtaaaac 60
ataagttcgt gacaaaccta atgtgaatgc aaatgcatat g 101
<210> 140
<211> 101
<212> ДНК
<213> Zea mays
<400> 140
ttcgcccttg atcatatggt gtgtaacttt catctgcatt aggtagactt yggtgttgtg 60
gttgtacgaa gtcgtcacta agcagcggag gatgtgctcg a 101
<210> 141
<211> 101
<212> ДНК
<213> Zea mays
<400> 141
gtttgaaagt gttgcgactg gcgactccaa agagtccaag ctaattaggc yaccacacca 60
catcgacgac gacgatgatc ggtgggtcgt cagtatggat c 101
<210> 142
<211> 101
<212> ДНК
<213> Zea mays
<400> 142
tggtgtgaac attcaaatca ccttgtctga actcccccca aattggtcgt yttcccgtcc 60
cggccactat aggtccatag ttgtgttcac cccccccccc t 101
<210> 143
<211> 101
<212> ДНК
<213> Zea mays
<400> 143
gtgtcgatgg ggagaggtaa gagctccttt tttgcatctc tgttcggctg yaagaagcag 60
gtctccgccg gcgggaggca ggaggaatcg gcggcggcga g 101
<210> 144
<211> 101
<212> ДНК
<213> Zea mays
<400> 144
tttgtcgctg cttcaactat tatccaactg tctctattct agttcttgcg ygcacaaaca 60
ctaggagagt gtaggcctct gcaacgagcg ttgtccaaga g 101
<210> 145
<211> 101
<212> ДНК
<213> Zea mays
<400> 145
cgccccttcg ccatgattta tactcgcgtg acgcccacac aacgcccatg wttagaagtc 60
gcccggccgc ccgcgcgctg ccgagatcca tcagctctta t 101
<210> 146
<211> 101
<212> ДНК
<213> Zea mays
<400> 146
tcccacaccc agattgttcc tttcaatttg cttgcttgcg tatttctccc kctccgatga 60
tcgatgattg cattactgat tgttttatgc ataataataa t 101
<210> 147
<211> 101
<212> ДНК
<213> Zea mays
<400> 147
acgaacggga aaattcctgt cctaacccgg tcagtattcc cacttttttc ygaccatttt 60
cgaattcccg gaaaaatatg aaaataggat gagaccaacc a 101
<210> 148
<211> 101
<212> ДНК
<213> Zea mays
<400> 148
cggtcttcat aggtgagaac atgtgcaggc ttctcgtgag gaactcgttg rtggtactcc 60
atagcatcag caactaatca tacaatacaa tattatgaca t 101
<210> 149
<211> 101
<212> ДНК
<213> Zea mays
<400> 149
ttcaccaaca gtgtccggcg tcggcgtgcc gaggctgtca aggcgcttct saggaatggt 60
ttgatcgggg gccagtcttg ggtggtcctg ctgcctcatg c 101
<210> 150
<211> 101
<212> ДНК
<213> Zea mays
<400> 150
gccccgccgg agaggatgac gcggcggtgc tcgcactgca gcaacaacgg ycacaactcg 60
cgtacctgcc ccgtccgctc cgccggcggc ggcagcggcg g 101
<210> 151
<211> 101
<212> ДНК
<213> Zea mays
<400> 151
ggccgacact aatcaaatca gctaaatatc ttttcccacc tgattaagtt rtatgggcat 60
gtgctgctgt gcatcacaat accttgatag atcacgtccc a 101
<210> 152
<211> 101
<212> ДНК
<213> Zea mays
<400> 152
ttccactaac tctttggcct ccaagtgttt cccacgtgga ggaagcagcc rgaacccatg 60
aaatcctaaa accaactcct ttgaatggta agggggtggt t 101
<210> 153
<211> 101
<212> ДНК
<213> Zea mays
<400> 153
tcagatgtgt tgtgccagta cccacgattt cagaggaaga agccggtgcc sggatcgcca 60
atgcaaagca aaatatgaca gcatcgagat cacgattcgg c 101
<210> 154
<211> 101
<212> ДНК
<213> Zea mays
<400> 154
acagtcccgc ttcaatcctg cccaccagct ccgaggttct ttctcgcccc raaaccgtcg 60
ctccccggcc gcgctgaaat catcgtgccc gggctctcct c 101
<210> 155
<211> 101
<212> ДНК
<213> Zea mays
<400> 155
gtcccgacgc tcctccacta gagcagaatt aaaacctcta tatatatgta sagcgagcac 60
gcctgtgcga acatgccgct ccagcacaca cagcacccat t 101
<210> 156
<211> 101
<212> ДНК
<213> Zea mays
<400> 156
gcgctagctg gctagtcctt ggtgataacg tatatcgcgt agatgatccc sgggagatag 60
gcgaggaagg tgagcaggag gcagagccag aattcgacct g 101
<210> 157
<211> 101
<212> ДНК
<213> Zea mays
<400> 157
ccggtggtcg ttggggttgg cttttgtacc tgtgcagaaa acagctcagg saggcgagga 60
ctcctagcgg aaaacatgac ggccgatgac gcgatgcgac a 101
<210> 158
<211> 101
<212> ДНК
<213> Zea mays
<400> 158
gtcacctcca tgaaacttcg gcaggaatcg cccggcaccc gccgatctgc mtcatcgatc 60
ctcaaatcag cagctgttct ttttcagtca tattgcgagc t 101
<210> 159
<211> 101
<212> ДНК
<213> Zea mays
<400> 159
aaaggagcta gtaaagccaa tcaattatgg atttgatatt gggtgagcga saggaggcgg 60
cagccagcag gcagggctcc cttatcctcc gtgatcttcc a 101
<210> 160
<211> 101
<212> ДНК
<213> Zea mays
<400> 160
tcctgcaaca tagttccctc ctaggaacag cccatcgtag ccacctcggt ycagggcagc 60
ttttgcggct tccagaagat caagatgtcc taccaggaac t 101
<210> 161
<211> 101
<212> ДНК
<213> Zea mays
<400> 161
gtccccaaaa accaagtcat ccttcagtcc gctgtccacc tgcgcgcgcg yataaaatcc 60
attcagcatc tcaattccat ggctccgcat gagtctgaga a 101
<210> 162
<211> 101
<212> ДНК
<213> Zea mays
<400> 162
acgttacagg tgggctggga atcgcattcc cgtgcgtgat tggttccgtt ratatccaat 60
ccctctcaaa gtctacacaa gacctgcttc gcctgacggg c 101
<210> 163
<211> 101
<212> ДНК
<213> Zea mays
<400> 163
tgagagagtt gatggagaaa cagaaactgg tggcatcgca gtagcggaaa wggtgctgcc 60
acttgactca ttctggttaa tccagagtaa attgttgcta c 101
<210> 164
<211> 101
<212> ДНК
<213> Zea mays
<400> 164
attacggagg aactatatgg gctcctacag tggcgtgtga cagggtggag yttgacggag 60
aacagcagca tctagtggct actgatatca agatttggtg g 101
<210> 165
<211> 101
<212> ДНК
<213> Zea mays
<400> 165
atatgctttc tttttctagg ttgaagaaga tgtacgtact ttaagactat ytcaagtgca 60
gcatttcaga taaaaattta gtcccgtctt ttcattcttg c 101
<210> 166
<211> 101
<212> ДНК
<213> Zea mays
<400> 166
gcctgcaggg tcttcctctt ctgttggcgc caggactcgt cgtcctttcc ygtcccagca 60
ccattgccgg agcagaccga tgaggtggcg gtggcgcccg c 101
<210> 167
<211> 101
<212> ДНК
<213> Zea mays
<400> 167
gcttgccaca gtctcttcct tgtactcttc ttcctctcct agctaggagt kgagttgggc 60
atacagtggc aattattttt cgttgcgcag ttggctgatc c 101
<210> 168
<211> 101
<212> ДНК
<213> Zea mays
<400> 168
ctcgcaacgg ccgacgatgg atgacgacga tagcactcgc cgaggacact sccaagagcc 60
tgcctccact ggcgctgcca ctgccactga taaagaaagg t 101
<210> 169
<211> 101
<212> ДНК
<213> Zea mays
<400> 169
ctcgcgatcc gtcgaagaat ccggccagga cgaagcggta tcgcctgacg scgaccggcg 60
gctacgtttc gcgctcctgg aggccgcggg agggccatcg a 101
<210> 170
<211> 101
<212> ДНК
<213> Zea mays
<400> 170
gaccactcgc cggccagtac ggaagcaggg acgccgacat ggagaagctt racgaggcgc 60
tgggcgtgcg tcgcgagcag acaggagagc gccaaggagc a 101
<210> 171
<211> 101
<212> ДНК
<213> Zea mays
<400> 171
cgctcaggga ggtacgccga cgaggagaac caaggacgca gaacgacgcc rtgggaggag 60
ccgctcggcg agctggagga gtaccgcacg cagggaattt g 101
<210> 172
<211> 101
<212> ДНК
<213> Zea mays
<400> 172
tcttcaagtg cggcttggtg ttcttcacgt gagcgatgca cccaaatgtg yggaagaagc 60
tcactactgg ctgctcgcca tgccaggcct cgtatggcgt c 101
<210> 173
<211> 101
<212> ДНК
<213> Zea mays
<400> 173
tcgaccgagc aatataatcg ttgccgcggg atatttattg agaaaccaac raatgcagac 60
acgctgcctg gtcgtcgtct acttgggtgc gaagtttgcc t 101
<210> 174
<211> 101
<212> ДНК
<213> Zea mays
<400> 174
gcttcgagga tcctgttgga gcctgcgtcg gagaggacgg gtatgattac rgcagggaga 60
cttcgtggaa cacgcctgcc ggtactgtgg gatccataat c 101
<210> 175
<211> 101
<212> ДНК
<213> Zea mays
<400> 175
ttataatctg gtcgtcgcgc agaagcttgc tagctagctg cctcctgtct ytgtctttgt 60
ctgcatcgat cctctatgcg ccgaaaaagc gtgatgtcga a 101
<210> 176
<211> 101
<212> ДНК
<213> Zea mays
<400> 176
tatgccctgt cagttgcttc cctccaccaa caaaacccgg caaccaaaca mtataccgtc 60
gtttttgctc cgcaagtctt ctctgcgcag cttggctctc t 101
<210> 177
<211> 101
<212> ДНК
<213> Zea mays
<400> 177
ttttgtcaac gagtgctccg gccgatcggc ttgtccgtac gcacgcgagt ygcgtcggtc 60
cggccgagcc gatgaggtcg ctcctcgctc gccccgcgtc a 101
<210> 178
<211> 101
<212> ДНК
<213> Zea mays
<400> 178
ttgggcagcg ggtggggtgt gcaaggcgac ccaactgctt tacaggtgtt stcattcatg 60
tgttgaatct acagaaaaaa aagaaagaaa gcgaaacatc t 101
<210> 179
<211> 101
<212> ДНК
<213> Zea mays
<400> 179
agttttgcag acagagccca atggatgtat cattattgac tatgattaac rgtggaaata 60
gaagtgacaa attgcataaa gagagctgca ttgtgctaat g 101
<210> 180
<211> 101
<212> ДНК
<213> Zea mays
<400> 180
gacaatgagt gaccttagag cttgcaacca aggtgctatg aaaatgcagt rccactattt 60
catttgcagc agagccaggt ccgattttca attttgtgga a 101
<210> 181
<211> 101
<212> ДНК
<213> Zea mays
<400> 181
aaggagcgga gggcggaggg gttctgcgtg ggcggtgatt gaaccgcaga wtggggcggc 60
tgctgctcag cctgtggaga atgaggagag ttgaggggag a 101
<210> 182
<211> 101
<212> ДНК
<213> Zea mays
<400> 182
ctcaccggta agggtcggta ccaactgatt ctccagtgca tggatcagta ytggccggtc 60
gaaccgaccg acatcggagc taaaccgatc ggtactgatt g 101
<210> 183
<211> 101
<212> ДНК
<213> Zea mays
<400> 183
taggcgccta tgacgacgac gaggtatggg cccccgtggc agtcaagatg rtcgcagcca 60
tccgcggcgc agagaacagc tgcggtgcgg cagaagtaag g 101
<210> 184
<211> 101
<212> ДНК
<213> Zea mays
<400> 184
aggattggtc acttaacaac acgattgaac tgcaactcca ggcccggccc yagttcttca 60
cgggacaatg gcagctgccc tgcagagcag atatcgcgct c 101
<210> 185
<211> 101
<212> ДНК
<213> Zea mays
<400> 185
catttagacc tgcttctgtg taagtagatg tagatctgct tcagtgtcag matgagaatg 60
tttatttcat gagttgctgg gaccttaagc ataagtttac a 101
<210> 186
<211> 101
<212> ДНК
<213> Zea mays
<400> 186
attggacagc gctggatttt gaccgggcca tagttccatg acttaggtgg yttatgttta 60
cttttcctcc ccaataatct tgggtcgagc gatgttataa a 101
<210> 187
<211> 101
<212> ДНК
<213> Zea mays
<400> 187
gttcagcgcc gcacaaagtg tgctgataac aatggattta catcatctac ygaatctttg 60
cagcctcctt cggcgtatct catcaactgt gagttctcca g 101
<210> 188
<211> 101
<212> ДНК
<213> Zea mays
<400> 188
ccaaccgcgt attggtcgta gtacgcagta taatcaccaa gcaagagtag scctttggac 60
aagagcagcg cgatgccctg tgacagccca agcaatgcag c 101
<210> 189
<211> 101
<212> ДНК
<213> Zea mays
<400> 189
cctgcaaagg gagtggtagg aggtgcacgc caaagcatgg ccggtggcgc kcgccccgtg 60
cttggcttct cttccctgtc ctagggcgac ctcgagcacg g 101
<210> 190
<211> 101
<212> ДНК
<213> Zea mays
<400> 190
tttacggaga ataacaagct ccggcacgaa tttaaacaga tccgggcaca rcatggaaaa 60
agtgcaaata aaaaagaggc tcgtagaaaa aaaaatcatt t 101
<210> 191
<211> 101
<212> ДНК
<213> Zea mays
<400> 191
tcgctcctcc tcaacggaga gaccctccct cagctcttca taaccatcgt mcgctacgtg 60
ctcccctccg aggaccacac cgtgcagaag cttctcctcc t 101
<210> 192
<211> 101
<212> ДНК
<213> Zea mays
<400> 192
gcgccgcgcg cgaggtcgct ctctccgccg ccgctgccgc tgcgtggcgc ygcagcacaa 60
acggacagca gcgagtcgcg cgccgcggcg aggaagggcg g 101
<210> 193
<211> 101
<212> ДНК
<213> Zea mays
<400> 193
ctgccgctgc gtggcgctgc agcacaaacg gacagcagcg agtcgcgcgc ygcggcgagg 60
aagggcggcc gcggtatcag gcacctgctg tgcagggcct t 101
<210> 194
<211> 101
<212> ДНК
<213> Zea mays
<400> 194
gctaaaaatt tcagaactgc cgatcctggc cgtcgatcat ggatccgagc rccatcccgg 60
caccactttc cattaataaa atttcgaccg ttggagacag c 101
<210> 195
<211> 101
<212> ДНК
<213> Zea mays
<400> 195
aacgtcggca acaagaagcc acttgttcct caggcagcag caagaggcct kcgacccctt 60
gccgacgtta cgaatcttat gatcaaagac cgtgctgctc c 101
<210> 196
<211> 101
<212> ДНК
<213> Zea mays
<400> 196
agcaggggag gggagagatg cgtgttcgtc ttccttccca cccatccctg yttctagctc 60
ctcctccttc cccagcgggt ggccgcagca atttgcaagc a 101
<210> 197
<211> 101
<212> ДНК
<213> Zea mays
<400> 197
cctctccctt cgtttcctgc tctctgctcg caccgaatcc ctcgtctcct rgctccatct 60
cagcatttcg gatccccgaa gaacaaacaa actgccgcca a 101
<210> 198
<211> 1753
<212> ДНК
<213> Zea mays
<400> 198
cggtagtggt gtgggcttgt gggaggctgt agttatggta gtgccgtggt ctatattatt 60
ggcttaatag atagtataga tttagtatat atcgtagcaa ttagctgttt tatccgacca 120
ctcatatgtt gtacacgaca atgacatttg ccaatagaag tagcacttca cttgttgttg 180
cacatatcag gaacaaagtt ttattgcagt ttgacagata tgcatgagaa atacacaaaa 240
agatgagtgt agcacaaacg aatgataatc aaaagaatgg atgaaacaaa aaagcgcaac 300
cttatggctt tctgatctcc gatggttgat atatatgtgt tgtgctggca cgacctcttt 360
cttcgatttc cacggcatct ggcaggctta gtataagcgg tggttaacga cgctgcactt 420
cttcctgatc tctcccttgc ttcctttcgg catgggcagc ttgctgagct tgagcagcga 480
gtccgagaag tcctcgtccc atagcgtggc gttctcggcg tactccacga cgtggccgaa 540
cgcctccttg tgcgtgagca gcgtccagtc ggagtggaag gtgacgatct tggccaggtt 600
gttgtggtag aaggagttgt ccaggaagtc ggggatcttg cgcacccggc tggtgaaccc 660
cggcatgaac ctcgccacca cgtcgtagtc ctcgtcgcgg acgttgttgt ccacgggcgg 720
gttggacccc tgggagcact tgtagttgag caggtcgcgg tacgccgggg tgatctgctg 780
cgatggctcg gagaggcggc cggtgaagga cgagcagtgg ccctggccga tggagtgcgc 840
cccggtgagg atgacgagct cctccaccgt gaagttcttc ctggcgaagt tgtcgatgag 900
ctgctgcacg tcttggttgg agtccgggag ctccgcctgg gcctcctcgg ccttggagac 960
gaagccgtcc aggcggccgg cggggacggc gaagtggacg tggccgttgc tgaggacgct 1020
ggccgcgtcg cgggccgcgt agatgaggat gtccgagcag gagacgacgc cggggcacct 1080
cttctcgaca gcggccttga tctcctccag gaggtcgaag gcggcgaggc cgatgttcac 1140
cggcgcctcc ttctccggat gagggttgtc gtaggaagcg tccaggagga cagaaccatc 1200
gcatccctgc attgcatgga tgtccagttc aatcaatcaa tcaactaata tacggccaag 1260
tagtatgtac atgaatgcgc atgctgctgc tcttattacc ctgacgaagc agtcatggaa 1320
gagaaggcgg acgagagcgg cgccggtgcg gcggctggcc ttgagcgcct tgatcacgtg 1380
ccacttcacc acgttctcca cgcccttgca tttcttgctg tagtacccga cctccagctc 1440
gctggcctgc gacgtcggca cggtggcgag gagcaccgcc gcctggacag ccagaagcgc 1500
gcagaggact gccaccgaga gcttcatttt tggtctcctg gagcctggtc cgcgcggccg 1560
ggcgcctagc tgatgaagca agagacagat gctgctggct caggaacagg cactttgatg 1620
gtgtgcttag gagatgggcc tgcaggggta tatattgccg ggtttggcca gtagtttaac 1680
tgttaatgat tgaggttgcc gccgataatg agatgggaga gaatagggag ggaacgtggt 1740
cgccttgcgg cgt 1753
<210> 199
<211> 5374
<212> ДНК
<213> Zea mays
<400> 199
atggtctgga tgacttgtcg gatcaatgtg tgttaaatga tttcagaata tatatatagt 60
atatgtattt atatatatat agtatatgta tttagagccc tgagttctgt ttaactattg 120
gaagctacga ccacaccact gaccacgtgt gcagaccgca ccgaacactt tttgtctttt 180
tgtacctaac ttcttacgtc aaattataat acgtgttatg ctaatttgtg tatcagttta 240
tacacttatg gtatgcgcat gttccgggca tccgggccca cgagaagatt gtgttcccgc 300
aatctgggca gtcgtactag tccccacaca aatattggta ttacgatgat ttatgtttcc 360
gtttacgaaa cagtaatgga cgtgtcggtc tatggacctt tgcgtagatt tcttcccgca 420
gattacgtgt ctcgggccca ggattccatc cacttctcac ggtagttatt ccgctcacct 480
cacccgaaac gcacgaggcg atcgaaccct agcattatcg cccgctgtca ccttgttctt 540
ctccaccgca gagaccgagt cggcttcgct ccaccgtggc ggccgaaccc taggggatct 600
cgccgccgtc gacttcatcg tatccaacgc agagaccgag ctgctgtcgc cttcagcttc 660
tccaccgcag agaccgagac ccagccgccg tcgcattggt cttctccact gcagagagac 720
cggaaatcgg ctggtcctca ttcccctcca cacaccggtc agtcctccac tgccatctcc 780
ttcattttct ccgccgcatt ctcgttgtcg ccgtcatctt ctccaccgca gggagacttc 840
gtccttttcg cccaactcca tttgaaggaa gcgctaaata attacattgg aggagctgtt 900
atcagtaagt cctgaatccc taacattcta tttgatttta tcgttgttct acattttagt 960
tcagcaactt tcattgcaat tacacatagt tgcatccggt tattcagact gaccatcatt 1020
tagttattga tctgcatatc atatttacac aaattgtatc gtaaatcagt gtcaccttat 1080
aataactcaa ttgtttcata tatcgaaaaa caaatcagcg taaattcatt gttaatttag 1140
cgtaagtcat ttagggctag tacgcaagaa agaacccaga tgtcatattt ggtaacctaa 1200
tggtcatttg tggtttgtac gttgtcgtgg cttactacat taatttgttg ttgcatatgt 1260
gcagattttt tttggtccaa aaggttgtat ttatacgttt tatatgtata tgtgttgttt 1320
gttttggcag gaaaatcata catgaagaac gtagaccgac ctccgcctaa tccaaagcgc 1380
attataattc agtctactca agagtgcttt agtgtcgaca taccccgtgt tccggcgaac 1440
actgtcagtc acaaaccaat gcatgccggt gatgaggacg atgaatttat gcttgtattg 1500
aagaagcgta accttgctag tagtcctcca agaatgatga agaaggtcgt tacaaagcgg 1560
atgaaagcag tggatcccct ccgacaggtt agaattgatg ttcaatgcca atataccatt 1620
ttctgcgtga aattatgttg tactgagttg tcaattaaaa actgagctgt gattatgcag 1680
atgtaaatgt tttacattat tcattgttac atttttacag tagaggctta atgttagatg 1740
caatccccaa gatgttatag taagcatcca gatcttaaat gccagacaac gtcaagctat 1800
tgcacggaag gggttctcca gtatacttga tatgagagtt gatgtgcttg gcagtagatc 1860
acatttatct tggctgatgg agaagcttga tcataatgat atgacaattc gagctggacc 1920
agggaaggag ctaaaaatta caaaggaaac ggtgcacctt atattagggt taccaaatgc 1980
tggtggagga aagccattag gcattgacga ggctgttgct gcaaataatt taagggttga 2040
acttggttta tccaaagaag aattcggtgt tgcagctcta caagatcgtt taaggaaagg 2100
atttgacaat gatctaagca tcagatcctt tttctgatac tgttcaatag gttgttgttc 2160
cctactgcta gttggggaat aactaaccat gaggttcttt taactgaaga aatgggccgt 2220
ttccatgaga tggactggtg ccagctcata tttaatgatt tataccaagc ggccaagaag 2280
tggcatacta ggagcaatac caatgtgtct acaacaattt acggatgctc catcgtcatt 2340
cttgtaagca tatcaccttt ttttttgttt gttttatgta gacaaatttt ggtatgcaaa 2400
tatcatctgc attagattat gatttgttat gtgtcacaac gattcatgtt atgcttttgt 2460
agctgtacta tctggatcat cttcatgact cagctgcacc tcagaataag cgtggaactc 2520
cgcacataaa atattttgat aggaatatta ttcaagcatt gacaagagct gacaagggca 2580
agatacgcca aggacaagaa ccatttgggc attgttcggt aattttttta tgcgcatcta 2640
ttatattttt tatctattta ttttggctta tatatggtac atggatagag gtagtgtcgt 2700
tttaatgtcc aaatgaaact ccattttata tgtgtgtatc agtttgtact ccatcttaca 2760
cgtctttttt tagttccgaa gcagcacaga gacctgctac acagctgtac cacatgttca 2820
accacaattt aaggcaagct tttatctata acttttttgt cttaaaattt caacataatg 2880
ttcagttata ctatatacat ttatttaata atattcattt tacaacacca taacaacgga 2940
acaaacactt ttgatgtacc gcgtcctgac ccatgtggcc agcacaccat tcacatcgaa 3000
ctcccgctca tcagggattt gatatcaagc aagctgaacc agcttccatc ccgccaccgc 3060
ctaggattca tagagaagct atcacattat gatacggtgg ttgtcaaggc ttgttcagtg 3120
atcaaacaga ctcttcagca gattgttgag aaaacaatac aatctatctt atgtttttag 3180
cgagttaatt gatgaggtcc tctgtgctga gtcagaggac aatgaagatg gttataatgt 3240
gcagcaacca actaaaaaat cagatgatat tcttcaccat tcggcaggtt agtcagtttt 3300
atggtcttta cgttaatcta aacaattata caaacatttc caatatcatt atgataccat 3360
tattaaatcg atgtattttt ttgcttaaat gttacgtaaa gaaggatgta cgagtccggc 3420
cactcaaact gtaccggata tgtctactcc agcatcccag catgagacgc agttgacaga 3480
cacacaggta catgaattac tattaataca acaacatatt cgtgtactat tacaaagttg 3540
ccctgatgaa gttgacattg atccactggt tcatacaatt ataacgaaac agaccaaaac 3600
acctacaacc ccagtggtaa tgttttttta ttacttgtat tttccttgaa ggtttgtatt 3660
catcgttttc tttaaataac atataaccat tatatttttt tcagggtcaa catggtgacg 3720
ctggcaagaa catgccgacc aacacaaacg tcactcctca agcaacacca gaatgctcat 3780
tcaacatacc acatacagaa tcttgctcgc cgatatttga tgcgacgccg caaataaata 3840
tatgattaca ccccctcgtt gtttttctat ataattatta gtagtttact aaacatctaa 3900
tccatttata ccataattgt tagtctcccg gcagtgtcca cccggcggca caaacaaatc 3960
tccccgacag tggcccatgt tttgatcaga ctccatctga acatgtcagc tcagtaatat 4020
acattttaat tataattgcc aaaactatgc agagattgaa atctatattt catatacctt 4080
ttttaggttt ctgttgtaac cgatccagat ataaacactt ctcaggttga ctaggtttca 4140
cttaattcga atatgcaatg tgtgttagct ttacgtgact acttgtgtgg tacgcaatct 4200
gacaccagca ggtataaagt ccagacaaaa ttaaaatttg tttatatcct ggtagttgca 4260
cgcgacctca ctaattgtta tcaaaatttc cacaggaaca taatagatta tggtgaatat 4320
tgttcgatat gttctgatat atatgaatct tccgccgatg ggaagtgcct cgacaatgtg 4380
ttcatgcagt ctttcattca atgtgttgat gatgatgcca aaaatcagca gctttccatg 4440
agttccaaca gattaattct cgacgttaat gttggggtaa attatcataa catacctata 4500
ccatagtcat atacacaatg gtatcccaag tatattgact ccattcattt tgtttattgc 4560
agtcactact gaactttgag gaacaggaac gacacagccg aaacccacaa ccttttgatg 4620
aatctgtact gcacacactt ctcgacaata cattgcctga gacaaacgaa ttggataaat 4680
gcaaagcggt aacaatcgta ttacatttta caaacataat ttatgttaaa tggtacatat 4740
cacatcgtca tgcataacca tttacatttc tgttgatctt tatgcttttt tacatggtca 4800
gatcatgatt cccatggtaa gcagggggca ctagacattg tatgttgtca atacagtcaa 4860
acggtgcatt cacattttag attccaatcc ctatgggcca acacttggtg gtactacttg 4920
gaaggattac cattttgccc aaatggactt aggtggcagg aagttaccat gggctaaggt 4980
tattatgagt aggttaaaca aagccctaca acatgtacgg cccaggtcat gctttcccaa 5040
atttggcaac ttcgcaattg acttgtcccc aaactgtcct actatggcag caggatccaa 5100
tgattgtggg ttttatgtta tgggatacat tctcttttat gattgcgtcg aaggatctct 5160
ccggtcagac atagaagcaa tacgtacaaa tttatcagtt cattttatag ttggctcaaa 5220
gcaacatatg gtacacatgt atatctaaca caattttatt ggcttgcagg gcaacactag 5280
ggaggtacgg tctcttgcgc tgcattacat cacattccat cgcaagaaca aagccatatc 5340
gctgcttgaa gacatccaaa gattcaatgt ctaa 5374
<210> 200
<211> 2226
<212> ДНК
<213> Zea mays
<400> 200
tcatttcgca cccaatttaa gaacttgttg ctcactactt ttgatcgttc ttcccttagg 60
cttggcctta tctggtcttg ctaagtttac accttccata tgaaactcca gtttctacaa 120
atcaaagata ttaagtcagt tttcattttc acatactaat catatattat agcaatgtta 180
ttacctttct agcttccaca tccgtagcac tactatcaac cagacacata ccagtgttat 240
gttccaatgt taatatcggc atattacatt gatcatcgtt agtattgcat tcatttttat 300
tagaccccat tacaatttcc tatgaaataa cacaaaacca gtgaagacac aaatgttata 360
gagcagcata aataatacat tataaaagtt tgttgcatac ctcgttccca tcatctaaat 420
tgcgtaaatt atttctaact aagtcttctg tttcatacgc ttcgacctat atcatttaaa 480
ttacatacat ggatcattat cgacatttaa taattgtgta ttcataataa cacgatgtgc 540
gtacctgaat gtcatcgcca gcactggtgc cacaactctc actagtacat atatcaagct 600
ccacaagcat caataaccct aacaattcgt tcatcatctc tagggcttta tcattcccac 660
ctttagatag actcgcatgg tgtactacct tcattgcctt tttaagcaac atcttctgtc 720
tatatgatct agtttctcca tcttttcctt tcaaattcct atcgtctctt gaaaaagcca 780
catcttgtcg cgcggagtat gtgtatcgtt gcaaaatata ctcactcagg atcctttcaa 840
tttggagatg catgaatgcg cgtacaagat gaacacaaaa taggcctata attgattaac 900
catacaatat gtcatatatt agttgcaaag agtgttatat tcgtaataat taatcatttc 960
atatagaaat atccgtttaa tataacacac ctgtatgctc ccaatgcttg cactcacatg 1020
aatacttccc ggcatccaca tcagccctta ttttgaattg gtgttgtccc caaacaattt 1080
tatttgatat tgtagtatgt ttcaccaccc aatcatttac atcaccctct gtatcacggt 1140
ctattcggta tgcagttgca tattttacgg tctcctagaa ccttgtcatt acagctctag 1200
ctagtgtatg tcctcgcaac tcttatttca aacatataca atgtagttgt atccttttga 1260
ccctgcaaat tacacaatac agtattaata ccggtttgtt tttaatagca taaaacatat 1320
gttttttatt tttgtcactt accatgcatc ctagtgcctc tttggcctct ttcatcttcc 1380
tactatgcag gagtttcatc atttgctttg caaattgatg aagcggtgtg tttgcatcca 1440
cgtgggcact cttcaccagc ttgttcatgc tctcgcttcg ttgtgtagag accataagac 1500
cacaatagtc tcctttgaaa aatgcaggta cccaatcttt acgtatttca tacaatctga 1560
caagggtgtt gttctcgtgt agctagaaat cattgagcat catttgccag gcagcctcga 1620
acttcatttc attcaatgga tgatgtatta tagattggaa ccttgttttg caatccattt 1680
cttcaaatct tgcatacaat tcattcaagt ggggcatata cctgttttgc acatgccaca 1740
aacacaatcg atgtattatg tttgggaata ctctgcctag tgcgattggc atcgcagggt 1800
cttgatctga aatcgataaa tttacataac attgtgcaag tcagtatgat atcatatagt 1860
acgtgtttaa tcaatacgag ccatttatta taacatacct gtcaacatca ctcgtggtcc 1920
gtcacatccc atgcatgttt tgaacgtatt gaagacccac tcgaatgtat caactgtctc 1980
gtctcctagt aaagcaaacc caaatattgt gcattgtagg tggtggtttg aaccaacaaa 2040
catgcctaat ggcttatcat acatattagt cttatgtgta gtatcaaagg taactgcatc 2100
accaaaatca atgtactcgg cttgctggct agcatgggac caaaatatgc ttaaaatctt 2160
cccttctttg tccagttgaa agtcagaaaa aaattgtgga ttatctttct tgcataatga 2220
aaaaaa 2226
<210> 201
<211> 2202
<212> ДНК
<213> Zea mays
<400> 201
atgtcaatcg tccgatccca cccgcatacg ttgctccccg cctcgaggct cgccggaacg 60
gacgcctgga tgccgcggcc gcaatccagc gccgcgcaag gaggctgcgc ccacactaaa 120
atggagcata gggaggctcc gcccccccgc aacccggagg atgcgcggag gccggaggga 180
atgggggaat tcgtgttgag gtctatgcat cgacatcttc ttcaaggtat acgtaccatc 240
gccatcccaa tccctacccc cagattgagc cagtacccca caacaacact gttgaacatt 300
ttcggcggcc catgcaattt tcggtggccc acctttagcc gccaaaaatt atctgctatt 360
tttggcggcc tgacacagcc gccggaaata aggctatttt cggcggcctc tgacacagcc 420
gccgaaaata gccttacttt cggcggctgt gtcaaagggc caccgaaagt aacgctattt 480
tcggcggctg tgctaggggg gccgccgaaa atagtctttt taaaaccgac gtcccttctt 540
cttctctgtt ttcctctctc ctctcccaaa tcagcccacg cgcgcctttg tccgccgccc 600
gcacgcctgc gcgccgcccg cacgcccacc tgcgcgccgc cgcgccgcgc cacctgctgc 660
ccaagccgct gctcgccccg gccaccgccg cccgcgctgc cactcgcccg ccgcgccgcg 720
ctactcggcc gtcttgctgc ccgtcacttc gagccgccac tccgccgtcg tcgtcaagtc 780
gccgcatcga gaggtatttt ttgcatagtt tatttcttat tttcggcggt tgttatttag 840
ttgccgccga aattagtata tatttgtaaa tgtttgtttg atttgtgtgt ttgattagtg 900
ttattgttta atttgatttc attaatatat tagtggtata tgtggtttag gtattggcat 960
attagatcta gcaatgcttt gtgaccgacc aacccaagtg ttcaatgaca acggtaagcc 1020
gccacgtgcc ttgttttgtc ttacctctaa agacaagatc gaggtaatga aactggaata 1080
aactcatatt tgcaggaggt tcaaactgga gttgtcttga gcccaatcga tgtttataga 1140
agagggcata gagcgaaaaa cactgaaatc tccgatgagc tgtgtagtca agcggctgtt 1200
gagtgtatgg ttagttgttg ttaggattaa tgttttcttg tcatatagct ataaattaca 1260
tgtgtgctga accttcgtat tttattgcag gagacatatg ggcaggagat ggttaggaag 1320
tatggagaag actacgattg gcgaggagcg cctaccatcg acgctgaagt tgtgcattct 1380
attggaggaa aggcgcatga acggtattaa cttgtgaatt ttttcaataa tattggaact 1440
atgaccgaat agctaatttt attgttttca agatattcta tgtttaccgg tgtgatcgat 1500
tcgacggagt tgcgttctag ttgcggctct tcgtcgctgg cgggcagtgg tagtagcagc 1560
cgtagtcggc gctctgtatc tagcatggag gacgctatga gggagcagca agaaaagttt 1620
cgtgaagaga tgcggcaaca acaaatgaca ttcctccaac agcaatcaga gtacatggct 1680
gcttacaacg cacaggcgca acaagcaacg aacgtgagtg tcttatttat tctcaacacg 1740
ctcgatattg gttctataac taatatatta tatttgcaac agtcttggtt tccacagcag 1800
gcacagcagt aaccatttgt tttcccttag tttcagtcgc cgatgcctca gtgggaatta 1860
catgctccgc cacctccacc tccacctcag gtgcttcaat ttagttaaga cttgtcgttt 1920
gtatcaacct aatttcaaat ctttactaaa cttgattttg tagggatcca aggtcatggg 1980
ccacaacacg ccaccaccgg tcatatcagc accaggagga gcttatgtag gggagggagc 2040
tactccaaga gaggttagta tgagaaacaa tttgtttgaa tagtggtcaa acatgtaacc 2100
aatgcagtta ttaatgtctt cttttttctt tacaatgcaa acccgttaca cgacttcatc 2160
aacgagtttt tcgcttctgg aggtagtgga cacaactcca ac 2202
<210> 202
<211> 691
<212> ДНК
<213> Zea mays
<400> 202
gacggccgtt gattcggccg gaaggggaga ggtatgtaac tttatgttct tcattcttgt 60
tcatattatg tgttcaaaat cataatataa actaatactt tttatttatc acttggacag 120
gtcttggacg gttttggatt atgctggggg tcatcgtcgc ccccccaaca acatcctcgg 180
cctgctgtgc agggaacact tccctggact tgtggagtac gccggagtga cgggcccagc 240
cttcaccttc gaccactacg ccgtcgcccc cgatgcagta gaccgggacg gcagggaatt 300
caataacaag gcggagcggg tgaagcaaga gctgtgggta agtcttagta taatataaaa 360
tatgtcgcat tcgttgcaaa ttcttgaaat aatgtatgga tacatcgttt ttgtatgcag 420
gatttcttca gatgcgatgc tggatacgag gctagggcgg atgtggtggc caccacgtgc 480
tgtaagaagc tcgtcgtgga catgcactat gaggcccgca tccaggccat catcacctac 540
cacggctccg tccttgggga gaaggtgacc aaacctcaag cccgaacaat gtcgttgacc 600
agggagcagt acctgcaggt aaagtcatgc atgtttggta ccagtactcc atgcatgaat 660
tttattttat cttctgatat gcactttatg t 691
<210> 203
<211> 2554
<212> ДНК
<213> Zea mays
<400> 203
ttgtcagtgt gtgcggtgta tggcaggttt ttggttcggc aagtggggcc ctccggggag 60
gaatctcagt aacaaaccgc tcttctgaaa aggtcagcca tccccggtcc ggtccggtga 120
tgtcgtcgct gtcgctctgc tagcttgctg ccgatccccc cccccccccc cccccttctt 180
ctctctaccc ctccctccac ctcataaata cttagtttaa taaccttgca ctgccgcagt 240
agcccttaac tgctgctatc tatctctttt ctgaaggaaa aaaaaggttt gatactcctc 300
tacctagcta gtcctgcatg ccgctaatgt gcgtcttgcc tgtttatttg ttcttaataa 360
gggctgccta tctattatat tttgcacctg ttttgctgtg ttcttggtaa ctagcttaat 420
tccttcgcct acaatcgtca aatccccccc atcatcagtc agatgaactt ttgatcgaat 480
tgaagttgtt cttctaattc ggccccagca gcgcccatgc atctggtttt atttgctttc 540
tgttgggtat aatatgcaag accttttgtt gctagggcaa ggctgcaacc acatgcgtgt 600
actgaactca tgatgtaact catccttttt gtttgctcac agaatcacta ctctactgca 660
cttccttttc atccgatccg caatcttttt tttcttttac atgctttagt tttctctctt 720
tcttgattac aaacatgatt actggaactt tcttaggctg ccttcccctt ccttggatct 780
gctttagttt tcttttttgg gctaccgcgc gcggcttatt tgagtttatc acttgctgca 840
tatacataat atatatatac atgcatgcga tggcgttcat gttactcaac tacagatctg 900
tttctgttcg tgtgtttcag ttcagcgcgc agttaagcat agcaggacga ccacgacgat 960
gtatcacccg cagtgcgagc tcctgacgat ggcgcacgaa acgccggacc tggacgccgg 1020
ccagccgcac ctaaccgtct ccggcgtcgc cagcatcccg gcagagctga gcttccacct 1080
gctgcactcg ctcgacgccg cggcggcggt caatcccgtc acggcgccgc cgcagtccac 1140
catcgactac ttcctcggcg gcgccgatcc ccaccagcag gccatgcagt acgagccgct 1200
gccgcccgcc gcgggcggcc accaccagta caccatggac atgttccgcg actactgcga 1260
cggccactac cccaccgccg agccgtacat ccgcgggaca atgactggag ccctcgtgtt 1320
cggggccacc gacgacgacg actcggccgc tgcctacatg cccggggggc actttgagac 1380
ctccccgccg ccgccacgcg ccaccggccg cggcaggaag cggggcaggg cgctgggcgg 1440
cggcttccat gctgtgctgg ccaacggcgt cgagaagaag gagaagcagc gccggctgcg 1500
gctcaccgag aagtacaccg ccctcatgca cctcataccc aacgttacaa aggtcgtacc 1560
aaatcctcct cttatgttcg tccatcgttt caaattaagt taaaaaatta attcacggtt 1620
cttgttgttt attttttgcg cactgcagac tgatagggcg acggtgatct cggacgcgat 1680
cgagtacatc caggagctgg ggaggacggt ggaggagctg acgctgctgg tggagaagaa 1740
gcggcgccgg agggagctgc agggggacgt cgtggacgcg gcgccggctg cggtggttgc 1800
tgccgccggt gaggcggaga gctcggaggg cgaggtggct cctccgccgc cggccgtgcc 1860
gcggcagccg atccggagca cgtacatcca gcggcggagc aaggacacgt ccgtggacgt 1920
gcggatcgtg gaggaggacg tgaacatcaa gctcaccaag cgccggcgcg acgggtgcct 1980
cgcagccgcg tcgcgcgcgc tggatgacct ccgccttgac ctcgtccacc tctccggcgg 2040
caagatcggt gactgtcaaa tctacatgtt caacaccaag gtacatacga atacgatacg 2100
tagccattga tcgatctgta attctgtagc ctgacgattc cgaggtttct ggtgctaaaa 2160
aatgcatctt tttttctcag atgacaatgc tttctgtctt tgttcaccgc agattcacaa 2220
ggggtcttca gtgtttgcga gtgcagtggc cggtaggctg atggaagtgg tggacgagta 2280
ctaggctacc atgcacttga atttctagct agctctacgt accgcgctgc tatgaatcta 2340
gctatagcgt ttcttggatg aaagactagt tagttgttac cttctatctt tgcttcaatt 2400
aaatccgctt gctcgttaca gactgagttt gtttctaaat gtcaaggttg ttttggtcaa 2460
attgaataaa ttggcacact ggcctgtgag gttattatat atatttatgt gtttattact 2520
ggtctattaa tttgtcttat tattaatgta ttgc 2554
<210> 204
<211> 440
<212> ДНК
<213> Zea mays
<400> 204
gttgcgcatg ttactgtgtt atgcaggtcc atgaagatgg ctgtacttga cacgttcctt 60
ttcacatccg agtctgtcaa tgaaggctac cctgtgacca gaaatctgat gcagtgcctg 120
atgcatgcct ccctgaagat cctgacagca aggttgcttg tgagacctgc accaagacta 180
acaacatggt catgatcctt ggtgagatca ctaccaaaga caacgttgac catgagaaga 240
ttgtcacgga tacctgccgt gggattggct ttgtgtccaa tgatgtaggg ctggatgcag 300
acaactgcaa ggtgcttgtc aacattgaac agcagtcacc tgacattgca caaggtgctc 360
atggtcactt cgccaagcgt cctggggagt ttttttataa ggaaggcttt attaatttta 420
agaacattac atcgaggtga 440
<210> 205
<211> 6507
<212> ДНК
<213> Zea mays
<400> 205
tttagaagta aaaggttttc ttcctctcaa ccaaaacaag taaaagcttt acttcagtca 60
ggatgcaaat gcaatggagt tacaagctag gcaagttcac aggttttact ataaacagaa 120
ataagttaga catttagtat aagaaacatg tacattacaa gaaactagct tcgctcggcg 180
caaacatctt catcatacac tgatacaatc gggctggctc ttctgcagtc tatcaattgg 240
ggccccaacg tatattgtcc cagaggaaag gcttcataca accaacttgt ggcaggctct 300
tcatgatcaa tcccacacag acgcagacgg cagcaatgga gaccaaggaa agcatcgcag 360
tcctattaga cagaaaccta tttgtccggg gatggaatgc aagctgctgg gcctgctgga 420
cacactgtct gcagctcaat gtttggttga tgcttagttg ttttttctca acaccaaatg 480
tgatttgatc ggcaggcttc attcggctac catgctttcc gacagtatca attgtgctgg 540
ggatggtaac cgagacatga gctctggtca cccttctgtc aattttgtcc tgtaccattt 600
gaatatagga tatgttgcct ctctttgcag catattcctc aggagtgaag ccagtggcgt 660
cacgagcatt cttccacgct ttgattccca actgagacat ttaggcagaa agaagccaga 720
cagcaaatgt ttcagtgctt gcacaaattc tcaaaagtaa aagaaaatgt ttttaaggta 780
aatgaatttt tatcatacta cacctcatgc ggaactctga tgtgatatta tacaccctac 840
acgatctatt caacaatcaa tgtaatacac aaaccaatgt tttgatggaa gtcctttctt 900
tttcttgata tgtacataat gcatttcaga tgaaaatatt tcaggtgcag acttaggttc 960
taacaataaa tggtggtctt agtttttatt ttaactgttt tacactaaga atcggaaatc 1020
tgctactttg aaatttgaat gtagatgtgc ttttttttag ttaaagcagg ctacaacagg 1080
taagaacatg gcaatttaga gtagttacct gctgaggatc atcagttaaa gcatctaaaa 1140
caccaatagc atcactgatt gtggctgcaa tatgaagagg tgtgatattt gacagttcag 1200
tcatcgcagg agtgaacaag aactgaactg gagcaccatc tccaccgccc actggagcaa 1260
tatttgttgt ggtgtatctt aatagaaatt caactaaagg ctttgagcgt tttttgaccg 1320
cagtatgcaa tagattttct cccaaggcaa attcaattgg tgacaataca tcaatatcac 1380
cctggaacat ggtgttcaga agcttcctta gaacagcaca ccattcctga tcaactgcaa 1440
aggatagcag ccatctaaat cttgcaacag ggaagcggtc aggacaatat tgtcgctgct 1500
cagacgtagc tcgcacatgg ctcctttgaa gaagccatcc gatttcatgc aaaaaatgta 1560
atgctcgact gcgataaatc atcagatcat ctattctttt ggaggtttga tcaaatgaaa 1620
ctaaatccaa tccatgctcc aacatccgaa tctcagaaca tacatctttg tccgtgacaa 1680
caaagggaac agaaaggctg ctttgatcat agtcttcaat ctgaaaacgg agtcaagtct 1740
gtaatataca caaaaaatga ggatataaaa aatatacaaa cattgtacct ctatgaatcc 1800
tcttccactt gtacaaggaa aggaacaaga gaaggtcagg cattgagggc cttgctgcat 1860
agttgaatct tcaagtagca tttgtgtctc ttcttgaatt aaatattttt catcaaacac 1920
acaaagtaat ctataacaaa atcagaatgt gtcagttaaa agaactaaag aagtaagttt 1980
agaggagaca tttaaatgtg aaaaaagata catactttgt ggttggctga actatattga 2040
aacctttcac agagaatttt actgacgaag aacaatcaac tgcaattgga gttacacaca 2100
gtctctgatg cttgtcacca attaccggtt gccagggaga tgcaaacata agactaccta 2160
acaaaaataa ggaaagaaat acaaatgaag ctgatcctcg aatatatgaa taactactcc 2220
aaataagatg aacggaccat tgcaactcag tgttaggcag tcctgtaccc tagcatacaa 2280
ccatcctgtt ctccagaatc catcattgga taagctaata agattttcta tccatgaagc 2340
tgggtcatca ttaaactata atagaaaaga aaaataacag taagcacttt gcttctgcat 2400
catatactat aaagaaaaca tatacagatc taaattcact gttaatgtag tgcaatacta 2460
cctcatccca catccaatta gggaggtgaa ggtaaatagt tagaataaca caaccaggtc 2520
taatataact ttccatatca gttggataat gcgacaacca gtttaggatc tgtaaattca 2580
aaccatttaa taaaagaatt agcattagcg tattaaaata tccacattga caaagttaat 2640
agaaaaatat gattccagtt tgtaacatga taactgacct gttcccgtag atctacagga 2700
aaatcttttg gctcttttcc aaagagtttg aacacaattt tatctgttcg gctctgcaaa 2760
tgttgtgcat aagagatatt aagtcggaat caaacatcag aaatacatct gggagaatct 2820
atctacaaat tctagagtaa cttccacaaa attacactaa gattggaaaa cctgagtaac 2880
tacacttcac atccacattc caggtctatc tcccactttg gatgattatt ctttccctaa 2940
gtggtggagc gaggtggttc gtttgacgcc aaagggaagg cgtagggggc tgaactctct 3000
gatcatcctt gctgcctggg aaatttggaa gcaccgaaat aactgtgttt ttggtttcat 3060
atgttctagg cactccacat tccacgtcta tctcccgctc tggatgatta ttctttccct 3120
aagtggtgga gcaaggtggt tcgtttgacg ccaaagggaa ggcatagggg gctgaactct 3180
ctgatcattc ttgctgcctg ggaaatttgg aagcactgtg tgttttcgat aatggaagac 3240
cgtgtatatt tgatcttcta aggaagattg tagaagaatg caaaatgtgg tgcatggcta 3300
gagcttcaaa gctacaggag ttcctggttg ggtcgctaga tggtttagtt tgagttttct 3360
tgagggtgtt ctttttgttg gtcgtggttg aggactctcg tctgagttgt gggttttgtt 3420
gtgttccttt tgtaatcggt ctggccttgc cagacctctt gttttttctc tccttaaatg 3480
aaatgacacg caaatctcct gtgttgttcg agaaaaaaaa aactacactt caaactactt 3540
tcacaaagct acacttttaa aaaccatttc cataatatta tacttctatt gtcaaatagt 3600
atcattagac tacacatttg tggttaattt gtatcgaaaa acaacagttt ttaagagcta 3660
aaatgtgtag ttttgtgata ttgaatggca ataattgtgt attttctgtg atttgtctgg 3720
acaatagaaa tgcaattcca tgaagttgct tctcaaaata tagctgagac agttttttag 3780
attggtttgg aaaaagtgta tttttgcgat agtttgtcca agattggtgt agtttatgaa 3840
atttactaaa catttacctc ggcttcttca atgcatgaat cgttcaaatc aaagtcctgt 3900
actcgtcttt tcaccgcagg ctcttctgag aacacaagtt attcaaatca ttttatttat 3960
acctttgata tttgacatgc taaatagaat caagtaaacg ataaagtgct ttacctgcta 4020
atgattctct cctgacagga attgaccgta tggtttgctc atgcattata gagccatttg 4080
cagcattatt accatcattt gatgatattg aagttgcatt ctttagaaca tcttgatatg 4140
catgtgtgct agcaatagca gcaaggttct tcaaaaggtt aaccaaagaa ttaggatggt 4200
tgatttgctc agaagagcta ccagctgtca tccacacaag cgcacagtta gaattgctgt 4260
ctggattgca tgtcgtttga aatatttgta ttttaaattg gtaatcaaat tttgtataga 4320
acttacactc gagcccggaa agttgtttta acagaaggaa taaggtgcta cttagagact 4380
gatcttcatt catggaattc ccgttcacag caggttgcgg ttgaactttc cttctccttc 4440
cattatgttg tgccagacgt gatcggcagc tcttcttacc atcatcaaat tcgtgaagaa 4500
ggtgaaacct aaaatggcat acaaattatt tactttcttc agctgatgag caaatgaaag 4560
gaaacataaa aaatgctcgg tgtattaaaa tatgaatttc aagttttatg ctaaactcaa 4620
tatcatgata ggctgctaaa gttaagatgt atgtagaaga aagagcaatg tttggttttt 4680
tttcttctgc tatgttgtac gatgcaaaat gttggcctac aaaaatatga catgtccaac 4740
agataactat tgcggaaata tgtatgcgat aatgaatttg tggtcgaaca agaagagatt 4800
gagcctataa ctatgatgtc ataggctagg ggtagcacca attaaagaaa aacttgtcca 4860
acaccggttg agatggttca gacacgtaga acgaagacct ctaaaggcac tagtgcctag 4920
tgggatccta aggggatagc aatgggaaca gagacaagag aagaccaaag ttgacataaa 4980
aagaaacagt aaaaaagact tgaaaggatg gaatatataa agtttgcttt agtcttaaat 5040
aagagtgcat gaaaaacaac tatccaagtg gttgaacctt agacttgtgc tttcggttgg 5100
gtttcaactc tgagtctctg acctacacca acttatttat gaacggatcc tatcatggag 5160
caaaaaaaac taatgtgttc agcatttctc aaggttaacg atcagtaatt cgtttgtgtt 5220
gtgtgcctgt gacaccaagg taatgagcct ttttatctct tttatttccc cctttgggtt 5280
ttgtaccgga gaagtgtttc tttttctttc tccaatacag gtagggcaca ggactcttgc 5340
tggagaaaaa aaattcagca gctgtaatct cacaacgaaa ttggtgacca acctgctgca 5400
ctgctgacag aaccgatgct ctacattatt gatgcagacc actgttgtcc tggtatgcgc 5460
ttcacacacc ttgtgcctct tatggtagtc cctggcacca ctaagatcag cttggcagcc 5520
gtcaacctgg caagaaggag tggtgctcga attggcacct ttcctgggtc tcgtctcctc 5580
agtgttccgc cctctctgaa cacacaccac gtgactgttg tctccatctg ctaccgcaga 5640
attgctgcac ccaccgctgc cgtcctcctc tggtgagagc cgccgcctct tgtcaacgac 5700
gacgccgaaa tcgatctccc cctgacctcg gctcagctcc ctgctgatgg actcggatgg 5760
cgcggcggcc gctggtgtgg ccaggaacag gttggcgtcc cacctccagt cgttcaaatc 5820
ccagtcgagg ctcctgttct tgttcccacc ggacattctc gccgcaccgg cttggtcacc 5880
gatctctttc cacaggaaac cggcttccat ttcctcggcc ttctctttcg agggaaattg 5940
accctgctgt tggcacgtat gacgagcatc caaagatcgc tggattcaat tacatccccg 6000
actacagccg aaacaaacac agggaattag atacgaacac caacacgaac acaaagggac 6060
tgccaaccct gggttgggac ttgggacaaa atcctgaaac tactgaagag tacaagaacc 6120
aagaagtacc ttcgttcggc cccggcggct agaacgatac cgtggttggc aagcaacgcc 6180
cttgaacatg aaaggcgaga aatgattgaa gaaatggaga tggaacagta gctcatggag 6240
agagccgagt caagtattgg gtaccgtggg ttaggaccgg ttggggatgg ggctactact 6300
tttcctctca cggatcctgg aggctggagc ggcggaacag aggcggaagc cggaagggct 6360
cgcggtggaa cggggggcga agtatcctgg ggaggcagag ccctccccac cggcatattc 6420
acactcgaac caccaccgcc tctttttcat ctcccctgtc tcttgtctgg cgctcggaca 6480
gcgacgccac gaccaagggc cggttgg 6507
<210> 206
<211> 6531
<212> ДНК
<213> Zea mays
<400> 206
ggttatcatt aaacgtttgg gagctgccct ttctttcctg agcagggtta tcattacttg 60
catataagct ataaaggggg gaaacgacag caagaacatc atgagttata gcataaccat 120
catctatttg gtttctatgt tcgaagctac atatacatat taagatttat agattataaa 180
aagttatctt aaggaccagc tccttgcatt tacatctggt ccttgctatt tgtaacattt 240
ctccgcttag gtataagcaa ttatgtatgc catcaaatag aggaacagag actagcacct 300
ggattgctac acaggttgtg ccagctcgct ctgttcatat atgtcttagc agacgtatgc 360
aggatcccat accatcacct gctatgaaga ggctgccgag tgaagggcga cgagggacgc 420
ataggcgcca tccttgatgt tgatcaaggc atcgtgcttt cccttctcga tgataacccc 480
attcctcacg accgcaatca aatctgcatt ttgtattgtt gacagacggt gagcgacgat 540
cactgttgtc cggttcacca tcaccctgtc cagtgcgtcc tggacgatcc gctcggactc 600
agcatccagc gcactagtcg cttcgtccag aagcaggatc ctagggtcct tcacaattgc 660
acgggcgatc gccactcgct gcttctgccc tcctgacaac tgggctccac gctctccaac 720
cactgtgtca tatccctgca tttttcaggt agacacttcc ctcagcaacg gtttttggtg 780
cttgattgat tgcatagaaa agccacatat aatattcctt gacatcagat gaaagaaact 840
aacctgcagc gccgaactga taaacttgtg agcatttgcc aattctgcag cagatatgat 900
ctcagattct gttgcttgtc catcttttcc ataggcaatg tttgccctta ttgtgtcatt 960
aaacaaagct ggttcttgac taaccaggcc catttgttgc cttaaccacc taagctggaa 1020
cttctgtatg tccacgccat ctagtagtat atggcctaca tctgggtcat agaacctctg 1080
gagcaatgaa attgccgttg atttgccact accactctct ccaacgagtg caacagtctg 1140
caaacaaata ctagagttgt tatgctcgtg ataataaaca ctgttgctca atacaggaat 1200
tatacattcc ctaaaaaaca ggatttatga attagaaatc acttgccttt ccagcatgaa 1260
ttgtcaagca aaggtcccgg aatatctgaa catctggcct ggtaggatac ttgaagctaa 1320
catgctggaa tacgatgttt ccttgtagtg cctcaacagt tacccctgca tcctcacttg 1380
ggtctatcct tgattttcga tctacaattg caaatataga agatgcagct gattttgctt 1440
tggaagaatc tgatgtaagg gtacttgatt gcgacactcc aattgccgcc attgcaagag 1500
ctaggaaaac ctgcgtatag aaggacaaat ttttagagct tttcattgga aagattcatg 1560
atactcactc caaatcatct ttaaaaaagg aaaacacaaa ccatgttgat ttgacttcac 1620
atttatgtag tcctttcttc cttaatatga aagccagata aggttaaatt tatcagtact 1680
accatagagt acttaccctg aaaactttag gaaaggttgt cttcctgtcc tcaacaagcc 1740
gagcaccagc ataaaagcta gctgcatata ccccaaatag caagaaaaag gaaactccaa 1800
aaccgattcc acttataatc cctgtcctga ttcctgttct tagaggacct tcacatttct 1860
tcttgtacaa atccatgacc ttctcttcag ctgagaatga ggccactgtt cttatgctgc 1920
ctactgcatc attagccacc tggcttgcct cttcatacat catctgcatt cacaaataac 1980
tggatgaagt ctgatgcgat agacaataaa tttaaggaaa ggcagttcag tacaagagaa 2040
cagacctttg cgtctgcact gaaaccgtga ataaacttca tctggatcca tccatttagg 2100
ccaatgagtg gtattaaagc taagatgatg agggatagct cccagtttga cacaaatgca 2160
ataaccaaac cagcaactag tgtcgaagag ttttgtacaa ccaattgaag cgcatccccc 2220
acaaggcctc taacttttgc tgcatcagct gacaaccttg cacctattgc tccacttgag 2280
ttttctgggt gatcaaacca ctcgacctcc atatttacca ccttctcaaa tgtcatcagt 2340
cggatccttc taatcaacct gcaccctgct atggagaaaa ggtacgaact gacagggagt 2400
gacaagaagt acactgcacc aaacaccaag aacatggaag cccagaactg tgaatccctc 2460
cttaggagat gtggaggctc atagaacgct tttatcacat ttgacaggag tatcgcaaag 2520
atcgggaaga taactccact gatgactgag gcaatggagc ctagtataag gaccgggatc 2580
tctgccttgt tgagagatgc aagccggctg agaggcactt cctgtggcat ctcatcacac 2640
agtttgttag atgaaccatc ctgaatgtca atcccatgag gcataccgaa tgggactgag 2700
aatgaatggt ggctactgtt atcacgggac gacctcctgc tagctgactt gtttattgat 2760
atttgtttgc ctggacgagc attggcatca accttcccat tgttctgctg gtttgcttcc 2820
tgtagcctta tcagttggct ataagctcct tctggatctc ttagaagctc actgtgtgga 2880
cctggtccaa aatgttaaat ggttaagcaa agatgcattg agcgatcaaa gactggcaaa 2940
tgatgttttt tgctattgat gtttcacaat gtaccttttt caaccagtgt tccctgatga 3000
ataacagcaa tggtatcagc atttcgaaca gtgctcagac ggtgtgcgac tatgactgtc 3060
gtcctgtttg tcatgaccct gtcgagagct tcctgcacaa tcctttcaga ctcggcatcc 3120
agagcacttg tagcttcatc caatagtagg attcttggat ctttcagaat agcccttgca 3180
atcgcaattc tttgcttctg cccaccagaa agctgtgtcc catgctcacc aacggaagta 3240
tcaaaaccct gtgaaatatc agaaaaagaa gagatctcaa tgcatgctgc ataattatcc 3300
aagccaatgg aaattgtatt tttcttatat atttgaataa gggcacaacg tctcctgttt 3360
ctttctaaaa aaaaagagca aaaacaaaaa tctgaaccac tgagagaggt aaaccttgct 3420
gattgaacag cattctgacc tgaggcattt tatctatgaa tttggcagca ttggcaagct 3480
cagcagcagc tctgatttcc tgatccgtcg cgttgtcttt gccataagct atgttctcct 3540
ttatgctggc agcaaaaagg accggctcct ggctgacaag gccgatttta cttctgatcc 3600
acctcaactg gaactccttg agattcacac catctatcag aacatcgcca agctgagggt 3660
cataaaaccg ttcgatcagg ctgatgaccg tggatttacc gctcccgctc tggcctacca 3720
acgcgatcgt cgtgccacta ggtatggcga gggagaaacc cttgaatatc tgttcgttgg 3780
gccttgtcgg gtaggagaaa tagacatccc tgaactcgat gtcccctcga acgtcctcca 3840
gcttcctccc cgtggtgctg tacgcgtcga tttctggcgt tctgttgatc gtctcgaaca 3900
ttttgtgtgc ggcagcctgc ccacctgcaa acgccttcat gcttggagac gcctgaccta 3960
gagccctggt gaaatacgat aagagatgcc aggtatcagg taaagcatca aacgttaact 4020
gaaaaaaagc agtcaagata ttggagacag agtcacagac agagagtttg aggggcaagg 4080
gtgaggacag gcaaaagagc aaaatacatc ctcaatagct aaaaacaaat atattgtcaa 4140
cacaacagca ggcaaaagaa caaagtattt tgggttctgt tcagcagaga gataggagtt 4200
gttagttagg agcagtccaa tatactactc ctataacagt atagcttgag aaagaaggga 4260
cgggtccatg agtggcttcc taatagtgaa cgggtggaaa agtctgctaa gcaattttgc 4320
ggcggttttg ttttctgaaa ggccacctaa cgcgtccaaa tcatcataaa caggaagctt 4380
tatctgcctg caaccaccct ttgcagtaac aacttgtaag tacgcatgat ggacccgagt 4440
ggttgcatcc aaaaccgtta cctcgatcat gcgccagcta ccaaatatag tggtttcgga 4500
tattcccagg atcaccaaac aagaaaatga ctgaactaca gaacctactg ttgttactgt 4560
taagaagggg agccttgaac ttacagagaa ccagtgagga ctgcaaagat gacgttcatg 4620
accttggctc ccgtgtaccc cttctccagg atgagcttag cgccatacca aatccctagc 4680
gagtagccgc agaagaggag caccatgacc gtgcccattc caagcccagt ggcgaggccc 4740
tctcggacgc cggacttgta cgcgttcttc aaggacatgt tgtacttgtc caccgcccgt 4800
ttctcgccgg tgaaagacgc gacctgcagc agcaggagga ggtttcgtca gtcagtcatg 4860
gtcacacatg tgtgtactgt ttcgtttgtg tgctctcacc gttctgatgg aaccgatggt 4920
ctgctccacc accaccgacg attccgcgta ggccgcctgg cccagggacg ccatcttggt 4980
gacgacgttg gacatgaccg cgcccgccag gacgagcggc gggatggtcg ccatcatgac 5040
cagggtgagc agccagccct gcgcgaaggc gacgatgaag ccgccgaaga aggtcaccag 5100
cagctggacg aacttgccga ccttctcgcc catggcgtcc tggatgagca ccgtgtcgcc 5160
ggacatcctg cccacgacct cgccggtgct ggtgtacttg tcgaagaagg cgatctcctg 5220
cctgaggatc gtcttgaggt acaggttccg tatccgcgcc gcctgacgct cgccggtgat 5280
catccagcag gtgacctctg gtcggacggc gaacagttga gtgctcagtg ctcagtgtct 5340
ggccgcgtta cgcaagaggt ggaacttacg gacgaaggaa gcgacggcgg aggccatggc 5400
gaggtagacg aagtcgagcg agaccatgga cacccggctg acgacgtcgt ggacgctgag 5460
cgcgccacca aaggcgtcga tgaggttgcc gaagagcacg gtcatgaagg gcaaggcggc 5520
gccgttggcc acggccccga gcgcgcccag cagcatgagc gccacgtcgg cggagtccgc 5580
gaacgcgaac agcctgtgga acggcacccg cgtcgccgca gcgccggggg gctcccccgc 5640
cccgcgcccc ttggcgccat tgcccgggct ccgagccgag gggccggacg cggacgccgt 5700
gcctgccgcc ggctgcaggg catccgcctc ggcgggtctc caggactccg gcatggctgc 5760
cgccggcgct cacgaatccc ccctcccccg cgcgagatcg ggcagtgctg gtcggcgcag 5820
aacgcgcggt gtgcctcggc tccgctcgcc cttgccttcg tatggttgtt gcctacctcc 5880
tagctcctgc ataccaccag cagcgcctca ctcgccacct gccacggccc aataattgta 5940
cacacattca gcagcggatc catctcagtg cctcacagct agaaagaagc tcgcagtagt 6000
agaaatccgg ggagcagagc agcacaagag caacagcaaa tctgaatcta taccgccgac 6060
gaggaattcc accagcgcaa gaagaccgat ttgggagata aaggagggca ggtgggggat 6120
ggaggtaaga gtttggaaat ggatgcgagc ttggcgaaga agtggtgtgg ctggcctgga 6180
gaagggaaaa ccgagcaggg aaagagttgg cagctttcgc acttgcccgc cggatgcgga 6240
cgcagaggcc agcagtccag cacccgcacc gcaccgcacc accggggaaa gagagagaga 6300
gagaaaatgc aagcttgaga cggggaaaga gagagagagg agaacggcgc ctggtagtga 6360
ttaaaatcta gtaggagaca gcagtacggt ggtttcggta tccatggcgg gcgagcaagc 6420
atataaaaat gtgagaggct accagtacca ctaccatacc acgcgctact caccatggcg 6480
gcagtgtcac agggtgggat aaagcatccg tgctccgtct cccactcgcc g 6531
<210> 207
<211> 12500
<212> ДНК
<213> Zea mays
<400> 207
atctcctagg aaagagaaac aacgaagact cgaaacgagt acaccaacct ttctttctct 60
cctctcgctt ctgcggcgaa ttttcttgtc ccgtgccggc cgccgtgtct accctgcgcc 120
accgcccctt ccaacgcctg ccgtgaagac aaaaggtgag ctttttatcc cctcttccag 180
tttcagtact tccccaaagt tcttcgaagg cgtttatcgg gtgccgggga acgcctcacc 240
gtccgcatga agccccagcg tgctgcggtc cgtcacaacc gccgttgcga ccttttgtgc 300
gccgtggttt ccccacggcg gcagcaagtg gagagggaga gcctgtggct gcgtcgattg 360
cgacggatgt gagtactgta cgggcctagg ggaaggtttg tgtatgatta gtgcttattt 420
ccaaaggttt ggacctgagt caaaagtgct gtttcaattg cttgtgatct gggatgtgat 480
tgctggtagg tacctctcgt caagtgcaaa gaaggtctac cttaattctc agtctctatc 540
tccgattcat ctgtcggagc tagttctttg atcataagaa tacatcgaaa gtgaagcttg 600
ttttagttgt ccttacctaa caacgacgac aacaacaaag ccttttccca aataaattgg 660
ggaaggctcg aattaaaaac ccaaccgggc ggtatgtgat aactgttttc cacattccaa 720
gtcttctttt aatgtctctt cccatgtcaa aaacttcggt ctttctctct ctattcccat 780
tgctatcgtg ccttaggatc ccattacaca ttggtgctta tgaaggtctc cgttggacat 840
gtccgaacta tatcaaccaa tgttgaacaa gttttttctt caatttatgt catacctagc 900
ctattacgta tatcatcatt tcggactcaa ccctttgtat ggccataaat cccacgcaac 960
acacattttc gtaacactta ttctggctaa acgtgtcctt gcccacaatt tttatatcta 1020
ttctttgttt aaagtagcta aggaagtatt ttttttgtta atcaatcagt tctttgtttt 1080
tgtttccttc tgtttgttct gttatatact catattttac agcttcttcc tgtcttgtaa 1140
acgtggtagg cattgcgatc cgaaacaaac ccaaaggtca tcgttgtatt actgtgctag 1200
taaaggccct ctaaggccct tctttttcct ttttaatata acgatacgca gttctcctgc 1260
gctttcgaga aaaaaatgag tgatcgatac acaatttcta ctaggtccag tggaggatta 1320
ggccatgagc gagcgattat atatataatg tttttctaat aatgatggta cacacggcgc 1380
aaccttcata gaatgccagg ggtgcgggat gggggttcga acccccgctg gctcgaccag 1440
gccataggcc tatctgcatt gaacctacat tttggtgcgg aaaccttttt tgcttgtaat 1500
tagtatgagc atttttttgt ttaaagaaaa tcctaagaaa aagcagggta tgatttagct 1560
aactaatact gaatgcttgg ttggtatggc ttatagtggc tcttgcttgt ttaattagtg 1620
ttttctatgt aactttgtaa taggtatatt gttctttttg tgcatcctta aatatctagt 1680
gggttctaga tgtctgattt agtaggcgcc gttcgatctc atggttttga tgttttttaa 1740
gtcgtcgagt aggtaccgtt tcatggagtc taatcaatgt ccacccaaaa tctttggcaa 1800
ttacttagcg atagtctaat caatgtccgc ccaaaatctt cgacaattac tttagtgata 1860
taaaatcaaa gtgttccttt gcatgcaacc taggtccccg acgtgaattg tttctgctga 1920
tgtactgcat gcatgcatac atgcataact aatacgtggc aagaacgcgg ccatgcgagc 1980
aactttcggc gcgttattta ccttccacct ccctcctaga gttccttggt atctgtaaaa 2040
cgcctctgtg tgccttagaa aagattatgg agggagtatt ccatagcgag atttatgctt 2100
tggatcatga caagaggtga tgagcgtcat ctagtgcctt gttttgcatt aagcttcatt 2160
actcagaagt tagaacatat ggtattggat taatgttaat tgctatatat ttaattctaa 2220
ccatggtaag tttgcactta aggcattgca aaattcttca atgatctagg atttattaca 2280
cctaaaaata atgacatgtt ggactttcta accatgacta gtttggaatc tttgattttg 2340
tcattacttt tataaaaatt gcagagagtc gttttctagt tcttttctat attttaatgc 2400
gagtgatctt tgctagtttt ttggttctag tgcctgcttg acatcgaagt gatttcttag 2460
gtcttctccc ttctgttgct agtgcttcct gatatctagc aacatcaaca acgacgtctt 2520
ttagccccaa gcaagttggg gtagactaga gttaaaaacc caacagaacc cttcccttcc 2580
tgtgcacttc ctattcaatg ctaaatcttt agatataccc tatcctttca agtctctttt 2640
tactgtcttt tcccatgtca acgttggtct tcctcttcat ctcttctcat tgttatcgtg 2700
tcttaggatc ccactacgca cttaagcctc tggaggtctt ggttggacat ctccaaacca 2760
tctcaaccga tattggacaa acaattgatg ctacctgttg gtcactcgtt ctcaattgct 2820
atacacaata aaaaacaagg caacacaata ttaaaagaga caatcaacct tcgtcttcaa 2880
gcataattcc ttcgggataa ctccttcgaa gacgaagatt aaaactttac tgagtgaatc 2940
gtgatttatt attacaagta agtgaacata taaaatatac ttaagtttaa acatgacaac 3000
ttgttttata tatcaataac atgtttcttc tccttcatac ataaagttta cgaacatacc 3060
gtcggctcta ggaaatgttt tagagcgaaa gtgaagtgag gagaaggtta cactcaacaa 3120
ttgcttggcc ctccaaaaag gtacttcatg cagtgcactg ttcatctatt tatagggaat 3180
catacaactt cgtacaatat tacatttatg ccctcaaaga ttacacatag ggtttataaa 3240
taatacaagg acgacattgt cttttgcctt cagcgtttag gatgacacct tctccttgca 3300
tcatcttctt gtgttgttat gacgaagcta ccttcgttac ccttccaccg ttccgtatcg 3360
aagctattct agtttcgaag ctcctgtaat acttggcaat atgctgatag caaatgatta 3420
gccgtgtttt tgaggacctt cggaagatga aggcccccaa cactaccttt agcctatcac 3480
gcatatcatc attctggaat ctggactcga tcacttcttg tatggccaca aatctaacgc 3540
aacatacgta tttctgcaac acttatctgc tgatggaagt aatcttagac aaagtggaat 3600
cagattttac agattcttga tagttgatat taaaccgtta aacatttatc agctgcaggt 3660
ccatgaaact ttcttctcgt gttatttgga aattctttgt tgatattgtg ggaaaccctg 3720
accctaacaa ggggttgggt gatgtatgtc atgtacgggc gctcatacgc caggcagcgt 3780
cgcgcccgtg acaggcgctg cgcccaggag cacgcagatc gccaggagcg cgatctagga 3840
gggcgcgtag atcgccagga tctgggagat ccgcaggagc atatggaaag tagtggctag 3900
gaaggaaaat cggaaaatgg aaagtagaag gaaagtagga attgattagt attactatgt 3960
ttaagcttgc ctaaaataaa ggagcgattg gctctaggac agcaggccaa tggccttata 4020
tgcggcacaa ggagagagga ataaaaccag aaaagaagtt atctcttctc cacctctctc 4080
taccaactaa gcctacggct gtgggggata accacgccgg agaagacgac gaaccgcggc 4140
tacgaccttc gtagccgagg gccccctgct ctagacgccc aggcccttga caacctggta 4200
tcacgagtca ggcgatcttc gtcctctcga tcccacacct ctgatcagtc ccccgtcgtc 4260
acccaaccac cacccagccc tccaccctca ccgccgtcgg cctcctccat tatgagcacc 4320
aaaaccatcg acgatctggc tgcgatgacc gagaagctca cgggcacgat ctctgtcctc 4380
caaaatgacg tcgagtcgtt gaagaaggac aaggcgcctt cctcctcgcc atccggcagc 4440
ggcggccacg acggccagca ccacatcgat cgaccgccga ggttccaaaa gatcgacttc 4500
ccgcgttttg acggcaagtc ggaccccatg ctcttcgtca accgctgcga gtcctacttt 4560
catcagcaac ggatcatgga ggagaaggtt tggatggctt cttaccacct cgagggcgtg 4620
gcccagtcgt ggtacctcca gctatagacg gatgagggga ccccgcgctg gcaccgcttc 4680
aaggacatcc ttcagttgcg cttcggacct cccctacact cggcaccgcc gttcgaactc 4740
gctgagtgta ggcggacggg caccgtggag gagtaccaga accgcttcca ggagctacta 4800
ccacgcgccg gaccactgga ggaacgccaa cacgtgcaac tcttcacggg cgggctgctg 4860
ccgccgctca gccatgcgat gcgcatccac aacccccagt cgctcgcggc agctatgagc 4920
ctcgcacggc aagtggagct gatggaactc gacagggccc cagcgccagc ccagaccagg 4980
ccagtcgcgc gcggcatcct gcctcctcct ccgccgcgac cggcgcctat ggcgcttccg 5040
gcgccccacg ttcctttgac gctccctgcg ccaccggcgg gcgcgactcc gggccgtggc 5100
gagggcagaa ggctcagcac tgaggagatg gccaaacgcc gtcgcctcgg attatgtttc 5160
aattgcaatg acaagtattc taggggtcac aacaggttct gcaagcgcat cttcttcgtc 5220
gaaggcgtcg agatcatgga cgaagacggc accgaccacc ccgacggccc cgacgctgag 5280
gcactgtgct tctcgttgca ggccatctcg ggctgctccg tggccgacac catgcaggtg 5340
gccgtcatgc taggggccac cccgctcgtc gccctcctcg actcgggcag cacacacaat 5400
ttcatatccg aggcagcggc gcggcgttcc ggccttcccc agcaacagcg cctgcgccta 5460
tcgaccatgg tggccaacgg caagcgcatc acttgcgtgg gcgtcatccg caacgcgccc 5520
ctcaccgtca gcggcgacgc attcccggcc gatctcttcg tcatgccgtt ggccgggtac 5580
gacgtcgtcc tcggcactcg ctggctaggc gctctggggc ccatcgtgtg ggatctcgcg 5640
tgacggcgca tgtcgttcca gcaccaggac cgtacagtct gctggaccgg cgtgcccacg 5700
tcctcggctc cggtgctggg cgccaccacc gcggccgagc ctcttctgga caccttcgcg 5760
accgtcttcg cagagccggc cgatctgccc ccgtcgcgcg cccatgacca ccacatcatt 5820
ctcaagcagg gcgcgcagtc ggtggaggtc cggccctacc ggtacccggc ggcacacaaa 5880
gacgagctgg aaggccagtg cgccgccatg atcgcgcagg ggatcgtccg tcgcagcgat 5940
tcaccgtttt cgtcgtcggt cctcctcgtg aaggagcccg acggatcgtg gcggttctgc 6000
gtcgactacc gcgcgctaaa cgcggtcacc atcaaggacg ccttcccgat cccggtggtc 6060
gacgagctgc tggacgagct tcatggtgcc cagttcttct ccaagctgga cctgcgttcc 6120
gggtaccatc aggtgcgcat gacgcccgga ggacatgcac aaaacgacat ttcatacaca 6180
cgacggcctg tacgagttcc tggtgatggc gttcgggctt tgcaacgcac cggcgacatt 6240
ccaggcgctc atgaacgatg tactccgtcc cttctagcgt cggtttgtcc ttgttttctt 6300
tgatgatatc ttgatatata gcaagaattg ggaggagcac ctgcaccacc tcaggatggt 6360
cctcgaggaa ctgcaacgcc accaactctt cgtcaagcgc tccaaatgcg ccttcggagc 6420
cccgtccgtc gcctacctcg gtcatgtgat ctcggcggtg ggcgtggcca tggacccggc 6480
gaaggtgcag gccatcttcg actggccggt gccgcgctcg gctcgggcgg tgcgcgggtt 6540
tcttggcctg gcgggctact accgcaagtt tgtgcccaac tacgacacgg tcgccgcacc 6600
gctcacagcc ctcctcacga aagacggctt cgcctaggat gacaaggcag cggcggcttt 6660
cggcgccctc aaggccgccg tcacctcagc accggtcctc tccatgccgg acttcacaaa 6720
gaccttcgtg gttgagtgtg atgcgtcgtc acacgggttt ggcgccgtgc tcacccaaga 6780
gggacacccg gtggcattct tcagccgccc catcgctcct cgtcaccggg cgttggcagc 6840
ctacgagcgc gagctcatcg acttagtcta ggcggtccgg cactggcggc cgtatctctg 6900
ggggcggtgc ttcgtcgtca agacggacca ctatagcctc aaatacctcc tcgaccagcg 6960
cctatccacg atcacgcaac accattgggt cgggaagttg ttgggctttg ctttcactgt 7020
ggagtacagg tcaggtgcca tgaacgtcgt tgcggatgct ctttcccgcc gcgacacgaa 7080
ggagagcgtg ctgctggcgc tctccgcgcc ccgcttcgac ttcatcgcgc gccttcgtca 7140
ggcccaggcc atggacccgg ctctagcagc catccatgaa ggagtcggcg tcggcacccg 7200
cgatgcaccg tgggtggtgg ctgacggcat ggtcacctac gacgggcgcc tgtacattcc 7260
cccggcgtcg cccctgctgc aggagatcgt ggccgccgtg catgacgatg ggcatgaggg 7320
agtccaccac acccttcacc gcctgcgccg ggacttccac ttccccaaca tgagacgcct 7380
ggtgcaggat ttcgtgcggg tgtgtacgac gtgccagcgg ttcaagtcgg aacatctaca 7440
tccagctggc ttgctgcagc cgctgccggt gccctcggcg gtctgggcgg acattgcgac 7500
ggaattcgtc gaagcgctac cccaggtgca cggcaagacg gtcatcctct tcgtcgtcga 7560
ccgcttcagc aaatactgcc atttcatccc cctagcgcac ccgtacacgg cggagtctgt 7620
ggcacaggct ttcttcaccg acatcgtccg cctccacggg ataccgcagt ccatcgtctc 7680
cgatcgcgac ccgttgttca cctcgtcgtt ctggagcgag ctgatgcgcc tcatgggcac 7740
caagctcctc atgtcgtcgg cgttccaccc gcagacggat ggccagacgg aggcggccaa 7800
ccatgtcatc gtcatgtacc tgcgctgcct cacgggggat cgtcctcgac attggcttcg 7860
ccggctcccc tgggcggagt acgtctacaa cacggcctac cagtcgtccc tccgcgacac 7920
ctcgttccgg gtggtctacg gtcgtgatcc gccctccatc cgctcctacg agcccgacga 7980
cacaagggtg gcggcggtgg cacaagaaat ggaagcgcgg gaagcctttc tggctgacgt 8040
tcgcttccgt ctcgagcagg ctcaggcgat ccaaaagctg cagtacgata agcaccaccg 8100
ggaggtcaca tatgaggttg gggactgggc cttccttcgc cttcgccaac gtgtggcagc 8160
gtccctgccg cgaaccatca ctgggaagct caagcctcgc tacgtgggac cctaccgcgt 8220
caccgagctc atcaatgacg tggctgttcg tctagaattg ccgtcaggcg cgcgcatgca 8280
cgacgtgttt catgtcggcg tgctccggaa gtacgtgggc tcgccaccca ccacaccgcc 8340
ggctctgccg cctctgctca atggtgcggt agtgcctgag cccgcaagaa tcgcgggggc 8400
tcgcttgtcg cgtggcgtgc ggcaagtcct cgttcactgg agcggcgaac cagcttcatc 8460
agccacctgg gaggagttcg acgacttccg cgcccaattc ccagccttcc agctcgagga 8520
caagctggcc ttcgacgagg gagagatgtc atgtacgggc gctcgtacgc caggcagcgt 8580
cgcgcccgtg acaggcgccg ccctcaggag cgcgcagatc gccaggagcg cgatctagga 8640
gggcgcacag gtcgccagga tctgggatat ccacaggagc atatggaaag tagtggctag 8700
gaaggaaaat aggaaaatgg aaagtagaag gaaggaaagt aggaattgat tagtgattga 8760
ttagtattac tatgtttaag cttgcctaaa ataaaggagc gattggctcc aggacagcag 8820
gccaatggcc ttatatgcgg cacagggaga ggaataaaac cagaaaagaa gttatctctt 8880
ctccacctct ctctaccaac taagcctacg gctgtggagg ataaccacgc cggagaagac 8940
gacgaaccgc ggctacgacc ttcgtagccg agggcccccg tgctccagac gcccaggccc 9000
ttgacaatgt attaatagac tgagttagtt gggtctggcc cattacatag gggtgagaag 9060
gtaaatacat ggggtaaacc ccaaacccta aaagggaact ctaacagata ttacttattc 9120
aaaatacatt actaattaca ttttgttgca atctttgttt attattttgg tattcaattt 9180
tttgagttat ggtcatgttt aggaagttga atatgtctgt acccggttac catgtctacc 9240
atagggaaac aatcatttgt aatttgaaca aatcctggta gtaggcagtg ctttgtttgt 9300
ttacacatta tctttctcca ttcagttact tgcagttttt agccttattt tttggctttt 9360
ttttagtttt tcctgcataa tgcttataag ataaaagtgt tttatttctc aggtcttggt 9420
taagtttgaa gatgtcgtgc ttggcatgct gtggcggcga agatactcaa agaacacctg 9480
ataatggagg tccataccct ggtggctacc caccaagtaa tgttcctcta aattatacta 9540
cctctgttct cgaatatttc tcatccgcta gtttattttt gaactaaacc gcgacaaata 9600
aaaaagaacg gagggagtat atgtaactta gcattttcta atactattat cgatattcat 9660
atatcagtct cataatccaa actattagta gctgacttta aaattgacct aaattatgtc 9720
aaccttcagg ggatgatgct tatcgcacag ctgatccaac tccgaggggt gctcaacctt 9780
tgaaaatgca gccaatcact gtccccacta ttcctgtaga agaaattagg gaggtcacag 9840
tggcttttgg tgatgaagct ttgattggtg aggggtcttt tggcagagta tattttggtg 9900
tactaaaaaa tggtaggagt gcagcgataa aaaagttaga ttcaagcaag cagccagagc 9960
aagagttttt ggcacaggtg tgcattgatt ttgacaatga attgtgcatc tttgctagtc 10020
tttcttctat aaaatatgtc tctacacctg caggtatcca tggtgtcaag gcttaagcat 10080
ggcaatgtcg tagagttgct tggttactgt gttgatggaa acacccgcat ccttgcatat 10140
gaatttgcta ctatgggttc tcttcacgat atgcttcatg gtattactct gtttacttct 10200
aaaatctagt gttctaataa cattgaaata ttttattctc tttagcttct ttctagatag 10260
ctgatgagtt tgaatttgaa ctccacagga cgcaaaggtg tgaaaggagc tcagcctggt 10320
ccagtcctgt cctggacaca acgagtgaag attgctgttg gagcagcaaa aggccttgag 10380
tatcttcatg agaaagcgca gcctcatatt atacacaggg acatcaagtc cagtaatgtt 10440
cttctatttg atgacgacgt atctaaaata gctgattttg atttgtcaaa ccaagctccg 10500
gatatggcag ctcgacttca ttcgaccaga gttcttggaa catttggcta tcatgcacct 10560
gagtatgtgc agttttcctt tcagtttcac tgacgcctgt atcctttgtt tgcttatatt 10620
tgaaaacttt ggtgtaggta tgcaatgact ggacaactta gctctaagag tgacgtttac 10680
agttttggag ttgttcttct ggagctcctg actggaagaa agcctgtgga tcatacatta 10740
ccaagaggac agcaaagtct tgtgacatgg gtaaacattt atattagcta ttatatgtta 10800
tagttctgtt agaggttatc ttgtatttgt gcaatactcc ctccggtgtc ctattaattg 10860
tcgttttgga caaggttcga gtcaaactta taaaattctg acaacaatta actattttgt 10920
tatttagttt tgaaacctaa tatttaaatg caccgatgtg tcttaaaaag tagttttata 10980
aaagtataaa tgtattaagg tagtgtttgg ttccagagcc tagttgaatg gaacgactct 11040
gttctagatt atgaggattt ctgtgtttgg tagttagaac agagccgctc tattttttgt 11100
ttggttctac agtgaagtag aatggagcgg ctccatcctg tgtttggtta caaagtgtag 11160
cagatcccta tggtattgaa ttatataaca agttaagaaa acttactgat gctattttat 11220
tatattaatt gtatatacaa acttacatat gctatacgaa ttaattaagc ttctacatca 11280
taactttata tacgaacatg catatactat atatatgaat accttaaagt ggctgcatac 11340
aagtcactcc acgaagtacg tacgagattt gctaatgccc gaagtaatgc aacgaagcag 11400
ctttgacgac gaaggaagag gacaggcgag cgcgggcgta cgcggcgcac acatccggtt 11460
gttttctggg agcgcacaca tgcatgatat tgagggaata ttcctatact agaacgactc 11520
cgttctattt ggatccaaac caaacatcca cattactgga acggagcggt tctatcctac 11580
caaactctag aaccaaacac tacctaaaag cttatttgta ttttaacaaa aaaacattgg 11640
tcaaaactat attttggaga ccgtgtcgtt gtcctaaaca acaattaaca ggacaccgga 11700
gggagtatat tgcattcttt taaaaaatac tcatgtaggc tttgtgctgt ttgacgaatc 11760
aagataatgc ctattttaat gcctgcaggc aactccacgg ctttgtgaag ataaagttag 11820
gcaatgcgtt gattcaagac ttggagtaga atatcctcct aaatccgttg caaaggtatg 11880
ataagttatt taggctctag ttgttgcgat ctttagattt tttcttagtg atatgttcct 11940
tcctcaaagg ccaccaaaag gagatacatg cttcatataa aatcatagac attaacattg 12000
ttagagcatc tccaacattg tcttaaactg gtgcctcaaa tctaaatata gagctccacg 12060
cagaaaaaac tattccaaca atgccttatt ttataaaatc ttggtaaaaa aattatagag 12120
caccctctca agtgcctcaa atatactaca ccgtagtgag ctgccctata atctagattt 12180
ggagatttac tgttagagca gaatatttta ttggtgccct aaattatata aaatatactc 12240
atttttaaat tatagggcat tttataggtc acgttgttgg agatgctctt atacttacat 12300
tatgcaaacc tgatattaaa ttatcagtaa cttataccat ttcatttatt gttgcttgtc 12360
gtcagttgat gttaaatgtt aatactccct gtgttccaaa atgtaagaca tagtaaaact 12420
atgtatctag acaacatata tttagatgca aatacaataa agccttttag tttcaagcaa 12480
cttgggtagg ctagagttga 12500
<210> 208
<211> 3507
<212> ДНК
<213> Zea mays
<400> 208
ttagtcgtat tcaaaatcag gatcaaattg ttttcttcga ttagatgtta tcttctttga 60
cttccgtttc cgttttttgg gacttactac atcttcagga tctcccacta atgattctgg 120
caaaacctca gtatctatat taaaattggc cggaagttca tcttcttggt aaatttcttc 180
aacttccgcc tcaaaccatt cacccttgtt gtgtaactgt tcacgaggat taactttgta 240
cactacccat gtggcttcca gctttataga tggatatggt atgtagtaca cctggtcaac 300
ttggtgagca agaacaaatt tgctacatgc acttgttttg agctcatgtt tgacttctac 360
catgccaaat tggttttcta cagtcccttg attgggatca aaccaatcac atttgaaaaa 420
caccacctta agatttttgc tcccagcgaa gtcgtactcg agtatgtcat taataactcc 480
ataataattc atctctcttc cacgatcatc gactgccctg catacaactc cagtattttt 540
tgtcgccgca aatggacgag tacattcaaa tttggtcgaa cgaaaacgaa accccttaat 600
gtcgtaacga ccgtagcttc tgacttttac gattgcgtaa gacatctgaa gcaagtcctc 660
gtctaaattt ggaattttgt cacactgcca atcagatggt gaaattagat ttctaaaaaa 720
atatataaag tagaacaaga gtgttaggtt atttacataa gctcgcaacc aattaatgaa 780
attgggcttc ccatgtcttc ctcgaagtgc gttttcatac tgcctcgagg acaatttctc 840
attaagttgc gattggaatt ctctgttaca acaagtaaac aaatgaggtg cggaacacga 900
ttactatata gatacacaaa tgacaaatta aatttcatct tacttgaaat atggggtcat 960
ctcatccata ttttcataca tatagagcaa agcgtccaac ttttcttcgc actcaggatg 1020
atattccgtg gatgctccaa cagtcttgcc ctcaatctca aaaatttgga gattgctgcg 1080
agtccgtact ttggcatcat gatacctcaa tgtaggggca ttgatattgt gctcgtctgc 1140
aaagtataca ctcgtgaagg ttgctacctc tttatatttg aattcttcag ctatacaccc 1200
ttcaactctt cctttgttac caaccattga actaagcttc tttaaagccc tttcaatatg 1260
gtacatccac ctgtattgca caggtccacc aaccttagct tcatatggaa ggtggacaag 1320
tagatgttgc attggattga agaatcctgg ggggaatatt ttttccaact tgcatataag 1380
gacaggaatt tctgtctcca acttctccat cacctctttc tttatttctt tagcacaaag 1440
ttgcctataa aagtagctta actcagctaa cgccttccag atactgactt tcaagtaccc 1500
acggaacatt atagggagga gcctttccat cattatatga taatcatggc ttttcaatcc 1560
agttattttc tttgtcttca agttcacagc tctcctaaac ccagccgcat aaccatctgg 1620
gaatttcaat tttttcaacc attccattac ttcatttttc tgtttaggtt ttagacaaaa 1680
ttgagcacgt ggcttttttc cattttcgtc gagttcctgg gttggtcggt tacaaagttt 1740
tgctaagtcc ttcctggcct ttatattgtc ttttgttttg tcgcctagat tcatgcaagt 1800
acttagaatg ctttcaccaa cattacgttc ctgatgcatg acgtcaatgt tatgcatcag 1860
aatcaatgcc ttaacataag ggagttccca taaaccacat ttatgtgtcc agttatgctc 1920
ggttccaaaa cctatgtatc catcaccact ttcattctct ttcaaattat taagcatagc 1980
ctcaatctct attccgctca gacgtctggg cggtcccttt gtcacgatag tgcccttctt 2040
aaaggtgtta acctcgaacc tatacgggtg tttttggggc aagaagcatc tgtggcagtc 2100
aaagtaacag atcttccctc caaactctag gcggaaacaa tctgtatcct tgccacatat 2160
tgggcatgtc agagttccat ggcaactcca tccagcaaat aaactgtatg ccatgaaatc 2220
atgaatggac cacaaatacg caactcgaag tgtgaatctc tcatttttgt agcaatcgta 2280
tgcttcaact cctttccata actttttgag atcatcaatc aagggttgca tcatcacatt 2340
caacccttta ccaggatggt ctggaccagg aataattagg gaaagaaaca tgtaatcata 2400
tttcatgcac agatgaggtg gaaggttgta tggaataaca aatacaggcc aacaagaata 2460
tgatgccgcg tttgtattga aaggtgtgaa tccatctgtc gccaacccaa ttcgaatgtt 2520
tctcgcttca ctagcaaatt ctggatcaaa gtcatctaga gccttccacg catcactgtc 2580
agacgggtgc gtcatcacat gagtgtcctc ccgttcacgt tctttatgcc atctcatgtg 2640
cctagctgtg ttcctcgaaa gaaacaaacg cttaacacga ggtgcaagag gcatgtaacg 2700
aagttgcttt tgggctaccg ttgatgtgac catttcacca tcatcatttt taacctctac 2760
atatctcgac ttgccacact ttagacactt actatcattc tcatgatcct tccagaacag 2820
catacaatta tctggacata catcaatttt ctgatagtcc atacctagac cagagagcag 2880
tttcctgcac tgatacacgt ctttcggcat cttgtgattt ggtggaaaca cttcgctgat 2940
taagttcaag agctcgttat aacaattgtt cgagaacacg aacttggact tgatagccat 3000
aagtcgagtc acgaaagcga ggactgaaac tgttgtgtgt tcgtgcaaag gctcttctgc 3060
agccttaagg aggtcgaaga actttttaac ctctggagga ggcggctcct catgatttgg 3120
tggaaactca aaatcaggat cctcccttaa atcttcaagc atctcgtcca ttctatcgca 3180
ctccacgtcg tcagtgttgt tggcttccgg cgaattttca cggggaaagt cctcgccgtg 3240
atacacccag acttcatagt caggcatgta gccatatttg caaaggtgtc ccgacagagt 3300
tctcttgtct tgacatctac aaatccgaca catactacat ggacaacgca catccgtgcc 3360
ggttcttgat atagcaaaag cacgatcaat aaaatcctct gtctttctta tccaatccac 3420
cgagggatca ttcatacgcc aaccttcata catccatcga cggtcctcac ccaccatatt 3480
tgatgctaaa atagtaaaac acatcat 3507
<210> 209
<211> 2955
<212> ДНК
<213> Zea mays
<400> 209
tcagtaagaa acctctgaac caagatcgtc tactaacgca gcagaaaatg ttggaaccag 60
atcagctccg ttgatgactg tagtgataaa atgcactcct gactctgcca gtttccatgt 120
catacatgca gctgcagaac aaaacatttt ctaagaaaat atatactcct taatatttct 180
acatagttaa atggaattta agcttatcaa agattctagt atgatagaaa cattgaactt 240
aggttgcaaa acgtataaca gttccaatca taatatcagc taaggactta actactgtgt 300
actaacattt ggattttatg aatagagttc cagaaccaat cacaagttca caagcagtat 360
gtactatcta ctataccaac ctgcccctgt gagtataaaa acataagaca agcaagtata 420
tatggtaatg aaaagagaga gaaaatgcca tgcacacatc gttttcatgg tttgattgag 480
aaattaaggt tctaaaagag aaaaaaaagc atagcataga aacctggagc aaatgccaga 540
caagtagtag aggcaaactc cttctgttcc ctcagaacgt atgtcaacag agcagctgta 600
ccacctccca gtgaatgtcc aacaacctaa atagatttgc taattgccat gaacacccat 660
caattgcaac actttgaaag aaacatgtaa acatagttat gatgcatcat aagtcataaa 720
aaggttatgc tgtagcaatg ctcatactct agcatcaaag agaaagaaaa tggctatgaa 780
ataactcaaa cctttatttt gaagtctggg tacatctgca atgcttgtgc cagacaaggc 840
cctgagagct ttgcaatcca cctagcagcg gcaaccatcc caatgggtct ccgcagcggc 900
aaccatccca atgggtctcc gagctgtagc aatgctcgta cttctccgca cgaccgctgg 960
ttcgtcccgt gggtctccgc gcgcgggggc ggggctgatt gggccgctgc ggagcgggat 1020
tctccgtccg cgtcccctac acgttgaggt tccatggcaa gaccaatgaa catctagtat 1080
tcgaatggta atggcctaag acaccaattt atcatattaa cctcagatgc ataacgtcaa 1140
ggatgtgagt tcatacagta catttctagc tcaaagaatt attctctaca ttacttttat 1200
ggaaagacat aagttaggtt aggttagtta tctagtcagt tcgtattagg aaaatctaga 1260
ggtcagtttc ctagttccct aagttagcta gatgacctct cttataaacg cagccgagtc 1320
tttgtttagg gatcaaccaa gaacaaatca aagttctcaa aagaccagca tggtcaaagc 1380
ctgctgccac agaaacaacc cctggttgat gagggaacct atcgctatta tcaatcataa 1440
caatattgaa tgtctaatag caataggaaa ctttatctat cttatatatc agtttcctat 1500
ttgcataagt tctgctgaca cctgtgatgg ttgtagtttt ttacccctta cttttaggtg 1560
caacaatcag tgctaaaaga catatagcat accttgccac actgagcaaa aaaagattcg 1620
acttcctcca gttttacatt gtaaggaagc ggtgatgcag caactgtcct agagtccact 1680
tgctctataa cctcatctgg cttcaacaac tccttagacc taccaacttt cttccctgtg 1740
gcaaagacaa tactgaacct taatacagga tataagcacc aagttactga tgtatacaac 1800
attccaacta aagtgagtat ttttttcatt tgaaccgtgc agaaaatgtc catattagta 1860
tgcgaccagg tgattcagtg atagttataa tgtaaggtcg aacattcatc agaattcagg 1920
aaagggccat tgttctgaca ttgctgaaaa tggaaatgga acattgccat attctgctga 1980
atgaaattaa gcttctacac catcttaatt caaaaatcaa ttttaactaa atactactac 2040
acgatttttg cataacacgt atggacgaaa agatagagat aacgagctta ccatcctctg 2100
atacgcggag cgctgtggaa tggcgcaaga tgtcggcaac gcaagtacgg tctcctctgg 2160
cactgtttct ggtttcgcat ccccttcgag acctaggtgt gacctcatcc acgagaagga 2220
acatatgaga gccaagctca ccactaacca atcagccccg cacccaaaaa ggagatagcc 2280
atgaattagc accatgaatt agcacctgga aaaatctcag agaaaatttt aaaaacatca 2340
aaatcaccat gaatgaaatg ctcaccagtg tttttcgcgg tggagatgaa ctggatgctt 2400
cgcctcgcga tgacgtagtt gccatgttgc tgctacgcga tccaaacgaa acccagccag 2460
ggatgaggaa ccgagactgc aatagaaatc aagcgtgatg gcgctaaaga aggtgaagaa 2520
gcaccagcct tctccatata cagacattcc cttccataca caagctacaa actctacagt 2580
cctgaaaaaa aaaacggtgc ccagccaccc aggagaaaat cacaaactca tgatcagata 2640
tacaggaaag gacagtcatg atctaaagct aaatccttgc aaagaaacat gatggattcc 2700
atgtacatac ctcttcgtat gtgtcgccac taactatact ttgttcagcc ccatctgaaa 2760
cattttttgc attagggtct tgtactcctg gcttcttgac tactcatcaa tctctctggc 2820
tcgcatcaca ggcaaagggt gtgaaagctc tctggtttgt gcgttcctgc aacatgtaaa 2880
gagattgaga aagaaatgtc atacaacaca aacacatatg catttccaca acaataatat 2940
cgtgaatcat ggttc 2955
<210> 210
<211> 836
<212> ДНК
<213> Zea mays
<400> 210
gtccagcccg gggttcctgg cgatgccgtt gaactcggcg ttgagcatcc ggatgatctc 60
ggagctctcg ttgttcacca ccgtcttcag ctgcttgtcc cacagcacct gaaacgcaat 120
ggaacactgg aatcagctcg gctgttgggc tactctgcac ttctgcgtgc tggaaaggag 180
gaggggggga atcagattcg ctgtttgttt ggtggatttg gagtgacggg attggaggct 240
cacggggatg gttggcttcc cggcgtagtt ggagctggca atctcgtaga gctccctcac 300
gctccaggcg ccgttgagag ggttgggctc ggcgccgggc tcctcgtcgg cgacggcggg 360
gaacacccag cccagatgct cgtcgctctc cttggtccgc tcgaagatgg gcttcatgac 420
ctacggttcc gatgcagcgc atcacgggga actggaatca atccactgcc gagcgaaccc 480
ccgatccggc ttgccggctc gcgatggtga gggcgaggct agggttaccg acgtaccgtg 540
acgccgatgg cgtggtcgag gcccttgagc ttgaggaagg cgaggcaccg ggatgcccag 600
gggcacgcgt acgagacgta gaggtggtac cgccctgcca cggtggggaa ccgcccgtcc 660
ctagaaacgg agctccggaa ggtggatggc gaccggtcga agtcgcccgt gtaggtgacc 720
tcgtccagcg ccgaccgcgc aatcatctga cgcgcgcgca tgagtccatg aacatagcaa 780
gcgtcaatca agagtccatg acggcagcat ctcggtgtcg tgggagggga tacgag 836
<210> 211
<211> 2054
<212> ДНК
<213> Zea mays
<400> 211
acaagagtta aaacgatcta atgtcgtgtt tgaagagtcc aataataaga ttcataaatg 60
tccatagatc aaccgaaact actaaactgt aattattacc caaatgcata acttaattag 120
ttattgctca gattaggcct ttctatacca tatatatgaa cagaagctga aacattgtag 180
gcgtcggtgc aattttcatc ttcatatagt aggccaacga cgtagataaa cacaccaaca 240
acacatatat aaatgtaaat ggacatgcgg tacagtaaaa acgtgtttca ttacagacta 300
gagtctacaa gaacgagtag tcagcagata acatgctaca gcagttcata ctttcatcaa 360
gaaattgaag tgagtgaaca aaggtaaaga cattgtaatt aagcgtaata acagtttcag 420
ttagaaataa ccttctcaga aggtctcagc ttggtgcagg agaaaccatc aagaacaagc 480
ttgcctacgg catctctcaa gacctcatac tcctccgtga cattgtaaac ctgatgtgag 540
attctcacgt accctgtcac ttgatcgccc ttggccatct cttgtccttc ctcaacactt 600
cttgagttgt tgaatattgg cacctcgacc ttgaattcgt tcctcagcat gtccctcact 660
ctcatcgcat catcatcgct ctcaatgcca aggcaacctg gcagcccgac catagccatg 720
cttccgcaca tttccggcgg cgagccaaga aacgtccccc aagcctcggc gagcatcgtg 780
cccatctcga tcaccttctc atggttccgt ttgcttatcg tctcgatgcc gccctcgaac 840
cgtctcatga agtcaacggc atcaggcaca acgagctggg cactgtaatc ccgcaccccg 900
atccaggcgc tctccatggg cagcccgttg ccgtactcgc tagacacgac ggggtggtgg 960
agctgcgagg ctacggggtc atccttgcgg atgtgcagga aggccacggc ggaagggcag 1020
aagaaccact tgtggaggtt gctggtgtag aagtcggcgc cgatgtcgcg cacgtcgatg 1080
ggcacctggc cgatggcgtg cgccccgtcg acgaagacct tgtccacgcc ctcctcgcgg 1140
cagatggcga cgagctcctt cacggggagg aggacgctgg gcatcgaggt gatgtggtcg 1200
atgacggcga ggcggactct gcggccccca gcctgggcga gcgcgagcgc ggcacggaac 1260
tcggcgacga cggcgaccgc cgaggcgacg gggaacggga gcggcacctc gacgacggtg 1320
gcccccgcgc ggaccacgta cgcgtggatg gacttcttga cggagctgta ggtgtagtgg 1380
agcatgagca ccacgtcgcc gcgtgcgaag gcgccctccg cgaagctcca ggccacgtgc 1440
tgcatgatga tggccgcggc cgtggtagcg ttgtcgacga gggagacctc ggaggcgtca 1500
cccgccccga cggcggacgc cacggcggca cgcgagcggg cgagccccgg ctggagagag 1560
tcgaagtaga acgcgtcggg ctgagagaga aaaagccgtt gccaccgcgc cctcgccgcg 1620
aggaccgacg ccgggcagca gccgaaggtg ccgttgttga cgcgggctac ggtgccgtcg 1680
tggtgcgcga actcggcgcg aatatcggct tccgtgatag ttgcgctggt gcgtttttcc 1740
ggcggcgcag cgttcctgcc cttggccgcg tcgtcgtgcg gagccgaggc catcggaatt 1800
tggggatgga gagggccggg tgcgggcctg aggctgagat agcgatgagg gggagagtaa 1860
tggttgactt gcaggagcca ggtgcccctg cccccttgat gtaaagaact ggtcaacggg 1920
ctaaggcatc tctcccgaag actttttagg gagcgttact gctccaattc aataaatttc 1980
tgctctaatt tgcaactctg tctggagcaa tcttcacgtt catgtggagt tagagctgag 2040
agaggcgttt ggct 2054
<210> 212
<211> 19
<212> ДНК
<213> Zea mays
<400> 212
ggcgtacttg cgcgcggac 19
<210> 213
<211> 13
<212> ДНК
<213> Zea mays
<400> 213
gttaaaaaat taa 13
<210> 214
<211> 11
<212> ДНК
<213> Zea mays
<400> 214
atgcattact t 11
<210> 215
<211> 20
<212> ДНК
<213> Zea mays
<400> 215
gtgctatact acctaaccta 20
<210> 216
<211> 10
<212> ДНК
<213> Zea mays
<400> 216
cccctgaacc 10
<210> 217
<211> 11942
<212> ДНК
<213> Zea mays
<220>
<221> misc_feature
<222> (1812)..(1911)
<223> n представляет собой a, c, g или t
<220>
<221> misc_feature
<222> (8603)..(8702)
<223> n представляет собой a, c, g или t
<400> 217
tttttttatt agattgtgga tacaagataa attattagct atggctttcg ttatgtacaa 60
taatatggaa accaacagct ttatatttag cttcaattgt actaaatccg ttctcgaata 120
tctgtcgccc ggttgatagt tcatttttga actaaaccgt gacgaatata ataaaaaaga 180
agactagcta gctagtagta tttacacgca tcgtcgtaat gaaacttaat ttataatcag 240
catatgatgg ctgcttaacg ctgatgagcc tactctttgc atatttcact cgcatgcact 300
tagaaataga gagacatgca tgcatgccag gtgtggttgg ttgtgcactc attcaaagga 360
gctaataatg gccatgttca aacgatctgt ataacgtttt tttttttgtg ttacgtatgc 420
atgcaagatc gatcttgtca gctgagaggc gcatgcgtac tgttactaat aataagatta 480
attatagaga gatcgattgc attatattgt tgcaaaacaa caagagatat gcatggtttt 540
acatgatgtg aactgtttat ttatttatgt aaagcaagga aatatctcaa ttattattgc 600
acaagtaaac tcttcacact actgtgcctg tggtaaataa atagtaattt tctcgtaaca 660
aacgagctac gtacgtctgt ctcgctagtt ggctacctac gtacgtttgg cacctcaaca 720
cacgttgcac tcaaattaac gaagaccata ctaacacctg cacctaaaca tcaacgaacc 780
aactcgtagg ggctgggcta attaaaatac atacaataca caagtccaag ctttttttat 840
gacgcctgtt tttttttcta gtaggatgta tttacactag ccggtgtatt aagtaaaccg 900
tcagtggaaa tatcatatac actggcggtt cacttaacta aaccgtcagg gtaaaacaaa 960
tatgtacact agcagttgta ttaggtgaac cgccaatgga aatatatata cactggcgga 1020
ctacttttgc gccaaaggtg tacactgatg ttccatggcc gagcgcaatg agtacatcaa 1080
caataggtac gagcaactgc atgatcccgt caaatatact cagtcgcaat gagtacatca 1140
acaataggcg tatttgtttg gttgcctgta tatctgaagt ataagttgca cgagtggatg 1200
caaaacaaat tattttaaga ctctttggct gcatccactc atacgccaaa aatactcgtt 1260
tctggtcagc ctggatcggg agagcgtggg tgcactcgtc catctaacca atcagccggt 1320
atatgatctg catctagcta tctaatctct cacatgtctc ccttgcacta ccggaatcca 1380
aggctttgcc gagtgttgca atctttgccg agtgcttttt gtcgggcact cggcaaagaa 1440
ggctttgccg agtgccgcac tcggtaaagc tagactctcg gcaaagaggc tctttaccga 1500
gtgctggaca ctcggcacag gacgacactc ggcaaagact agtttgccga gtgtcaaaca 1560
ctcggcaaag agggctctcg gcaaagggcc gtcaacggcc gtcctaaagt tgacggccgt 1620
cagtctttgc cgagagccag cggctggcac tcggcaaaga ggattctttg ccgagtgtca 1680
cacagctggc actcggcaaa gcttcctttg ccgagtgcca gctgcgtggc actcggcaaa 1740
gaataggggg gagtctttgc cgagtgtcac caacgggaca ctcggcaaaa acgaatcaag 1800
ctgatcatcg cnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 1860
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nttcggcaaa 1920
ggaggcctct tccgcctagg gccgtcaacg acgtcctaaa gttgacgccg tcagtttttt 1980
ccgagagcat ccgctgcact cgcaaaggag gattttttcc agtgtcacac cgtgactcgg 2040
caaagcttcc ctttccatcc agctgcctgg cactccgtca aagaataggg atagagtctt 2100
tgcgagtgtc accaacggac actcggcaaa gaatgtttcc agtgccagcg atctggcact 2160
cggcaaagct tattttaaaa ttaaaaaaaa aaactttgcg agtgcccaga tcacgggcac 2220
tcggcaaagc gcccctatta caacggccga atgcttcttc tcctcactct ctctctcact 2280
tactcactca aacgccgtca cccccgcgcc ctcaccgtcg ccgccgtgcc gtatcttcgt 2340
gccggccgcg cccgcccgcc gtgccccatc gcccgccgcc gccggccgcc cgtgcgccat 2400
cgccgccgcg ggtcccgccg ccgcgcccgt gcacgcgccg cctccgcgcc ctcgccgccg 2460
cgctcccgtg ccgccgcccg cccgcgtcgc cctcacgctc gccgaagccg cgtccgccgt 2520
gcccgcgctc gccgtgtcgt tgcccgcgct cgccgtgccg tgcccctcgt cgtgtcgcgc 2580
cctcgcccgt cgtcgtgccc ctcgcctaac ccctcgccct tgcccgtgcc cccgccgcgg 2640
ccccgcccac cgcccccgcc cgtgcccccg ccgcactcct cgtccgtcgt gaccccgccg 2700
tttccccgcc gtgccctcgc ccgccaggcg cctcgcccgc gacccgacgc ctcgcccgcg 2760
acccgtcgtc tcgcccgcgc cccgtcgcct cgcccgtgcc aaaacctcta aggtgattac 2820
ttgatttatg ttatacgtgt atgatttata tgaaatgatt agttttaata ttaagttagt 2880
tatatacgtg catgattagg ttaatatgta gtattgtgga ttgccggcca tcgtgccgtt 2940
gaattatgcg tggatgtcag ccatcgtgct gatagtttta tttgcaggat tttgaaacct 3000
ccccgtgcag gggaggtgct gccgaaattt tctgttgcta ggcttctgtt agaggatgga 3060
ggaccgtgag tggatgtaca tgggccgtag aggaaggaac gatgtcacca ctgaatggat 3120
tagaaagacc gatgatttcg tggaacgggc atatggcgaa gctgctaaag gggcgaagct 3180
agtcccatgc ccgtgcggca aatgtgacaa tcggaaaaga aaaccaaaga aggtcatggt 3240
agaacatatt tggaagaatg gatttacgcc gggctatact cggtggatat ttcatggtga 3300
agcgcatcgt acgagagagg aggtgttgag acaacgtgtc gaggattatg acgcggatgc 3360
gggggtagca gatatgttga acgactatca ggaggcacaa tacacgggag gatgtatgga 3420
tgacgagcca gagccgactg caaaggcgtt ctacgacatg ttcgacgcgg cacagaaacc 3480
ccttcacggc cagacaaagg tttctcaact ggatgccatt gggcgtgtaa tggcattcaa 3540
gtcgcagtac agcatgagta gagacgcatt cgatggcttg ttgacggtta ttgggagtct 3600
gcttccggat gatcacgtta tgccaaagag catgtacgag gcacaaaaac tacttcgtgc 3660
actcaagatg acgtatgagc agattcatgc ttgtccgaag ggctgcgtgc tatttaggaa 3720
agaatacgca gaggcaaagt actgtcccaa gtgtaattcg tctaggttta tggaggtaga 3780
ctctagtgat ggacagaaga ggcagctcga catccccttg acaatcctac gacaccttcc 3840
gttcataccg aggatccagc gtctttacat gacagaggaa tccgcgaaac agatgacatg 3900
gcacaaaaat ggaaaacgat acaatcctga caagatggtg cacgcatcag atgatgaagc 3960
atggaaacac tttgatgaca ttcaccgtga gaaagccgaa gaggctcgta atgtacgtgt 4020
tgcgttggcc acagatgggt tcaatcccta tggaatgagc gctgccccgt acacatgttg 4080
gcccgtgttt gttatcccaa tcaatctccc ccctggtgtg tgctttcaaa ggcagaatat 4140
attcgtgtcg ttgataattc caggacaccc gggcaacaaa atgggcgtgt atatggagcc 4200
tttgatcgat gaattggtac gtgcctggga ggaaggggta tggacgtatg accgagctac 4260
gaagacaaac ttcagaatgc atgtttggta ccagtactcc atgcatgact taccggcgta 4320
tgggctactc tgtgcctggt gtgttcacgg taagttccca tgcccagttt gcaaggaagc 4380
tcttaggttc atttggttga aaaagggtgg caaatattcg tccttcgata aacatcgaca 4440
atttcttcct gctgaccatc cattccgcct agacatcaag aactttacga aaggtgtcgt 4500
agtgacagac cgcccacctg caacgatgac tggtgccgaa attcgtcaac aaatagatgg 4560
gctcgtggcc aatacagaag gtggttttgt gggatatggt gagcagcata tgtggacaca 4620
taagtctggt ttgactcggc tcccctatta tgacgacctg ctccttccac acaacattga 4680
cgtgatgcac actgaaaaaa atgttgccga ggcactgtgg gcaacaatta tggacattcc 4740
tgataagtca aaggataatg tgaaggcaag agtggatctg gcagcgttat gtgatagacc 4800
aaaactagag atgaagccgc caagtggcgg aaagacatgg agaaggccta aggccgactt 4860
cgtcctaaga agggaccaga ggaatgaagt actttagtgg atcaaaattt tgatgttccc 4920
tgatgggtat gcagctaacc tgagtagggg ggtgaactta tctactatgc gagtcttagg 4980
gatgaagagt catgacttcc acatatggat tgaacggatt cttcctgcga tggttcgagg 5040
ctatgtccct gagcatgtct ggctaacgct ttcagagttg agctatttct tccgtcagct 5100
ttgtgcaaaa gagttatctc ggaccgtggt tgcagacttg gaaagattgg cccccgtgtt 5160
actgtgtaag cttgagaaga tatttccacc cggcttcttc aatccaatgc agcatttgat 5220
tcttcatctc ccctatgagg cacgaatggg gggccccgtg cagggacgtt ggtgctatcc 5280
aatcgagaga tgtctaaaga ctattcgaaa gaaatgtaga aataaatgca aaatcgaggc 5340
ttccattgca gaggcataca ttctagagga ggtctcaaac ttcacaacaa cttattatgg 5400
tgacaaactg ccgagcgtgc ataatccacc ccctcgttac aatgatggcg acaatgaatc 5460
gaaccttagc atatttcgag gccaactcga aagcgcaagt ggctcgacca cgaagaccct 5520
gacacatgaa gagtggcgac atatcatgct atacgtattg accaaccttg aagaggtgac 5580
gccatacatg gaacaatttc ttcatgaatt ctggcgtcga tcaagggacc ccactccaca 5640
ggaatatgac gctcttctta gaaagggtgc gagaaatggg ttgcccgatt tcatttcctg 5700
gttcaaacgt aaggtacgtg cttagtttgt aaaagagata cgcctttgaa ctcaatttag 5760
cgtcttttaa cttgcgaata cttgcagggc caaagagatc cgtctatgag tgccgagttg 5820
agacaagtag ccaacggctt tgcttatagg gtcaagaaat attctgggta tgacgtcaat 5880
ggataccgtt ttcgcacaac acactacgac caaagtcggc ccaatcggaa aacaacgtgt 5940
tctggagtct ttacgccggg gcttgacaat gtcgattatt ttggacgaat tgaagaaata 6000
tatgagctca atttttatgg ttccaaacct cttactccag tgatattcaa atgtcattgg 6060
tttgaccctc aagtgacgag acggacacat tctaatcttg ggatagtcga aattcgacaa 6120
gattccacct tagcaggaga cgatgtctat atcgtggccc aacaggccac acaagtgtat 6180
tatctcccat atgcgtgtca aacgaaagaa catcttaagg gttgggatgt tgtgtataag 6240
gtatcgccgc atgggaggtt acctgttcct aacgatgaag attacaactt agacccggac 6300
acatatgatg gagagttctt ccaagaagat gggctagaag ggcgatttga gatagactta 6360
acagaagcta tcggaatgaa cgtagatatt gaaatggttg ttgatgagga ggatgatgag 6420
gtgcaaaatg ataatgactt agtaatcctt gaaggcaatg atgaggttgc gtcttccgat 6480
ggtgttgaga ttgaaatgct tgatagtgat gatgagagtt ttgatccggc taaccccgac 6540
acatatgaag attattttta atcgatgtaa tgctatatga ctttttattt cgcacctgtt 6600
tttaaataca tctttttata tgtgcttatt tgtttactct taattgcagg ttgttggaca 6660
aatatggtgg gcggtttcct gaggaggagg agggggagga ggagtaggac ggcggacgat 6720
gcagagcagg cagcgcagca gcacgaggca gagcaggcag cgcagcagca ggcggcgcct 6780
caggacgacg acgaccagca gcaggacgac gacgaccagc agcaggacgc ctcaggttca 6840
ggcggctcta gttcgaggag catctacctg cgaggccccg cgagtctccc gccgcgtccc 6900
atacttcggg acagacggcc gttgattcgg ccggaagggg agaggtatgt aactttatgt 6960
tcttcattct tgttcatatt atgtgttcaa aatcataata taaactaata ctttttattt 7020
atcacttgga caggtcttgg acggttttgg attatgctgg gggtcatcgt cgccccccca 7080
acaacatcct cggcctgctg tgcagggaac acttccctgg acttgtggag tacgccggag 7140
tgacgggccc agccttcacc ttcgaccact acgccgtcgc ccccgatgca gtagaccggg 7200
acggcaggga attcaataac aaggcggagc gggtgaagca agagctgtgg gtaagtctta 7260
gtataatata aaatatgtcg cattcgttgc aaattcttga aataatgtat ggatacatcg 7320
tttttgtatg caggatttct tcagatgcga tgctggatac gaggctaggg cggatgtggt 7380
ggccaccacg tgctgtaaga agctcgtcgt ggacatgcac tatgaggccc gcatccaggc 7440
catcatcacc taccacggct ccgtccttgg ggagaaggtg accaaacctc aagcccgaac 7500
aatgtcgttg accagggagc agtacctgca ggtaaagtca tgcatgtttg gtaccagtac 7560
tccatgcatg aattttattt tatcttctga tatgcacttt atgtgtttac tttgcaggtg 7620
attccacatt ggtgcgccgc acatcctctc tgctgggagc agatggtgga taggtggtgt 7680
tcggctgagt gggacgaggc gcacacagct agccgggaac ggcgtttgca gatgcaaggc 7740
ccctcgcacc accaaggcag ccggagcctg ggccaatatg ccgaagcatg ggtacgccac 7800
cttttttatt tagatatata gcactaagtt agatattatt tctaactatc tcgttggttt 7860
tcgttgcagt cggcgtcaca tggtggcagg ccttgttcca ccttctcggc ctatgctatg 7920
gcccataagg gtaaggcgac gtccgacgtc acctacaacc cggatgacgg gcccgaggcc 7980
tacaccaacc ccgccgtcta cagccgcctc catgactaca ccgccatggc gcaggaggtc 8040
catggcccag actatgatcc gagcaccgag cctatcgacc ccgatgtgct catgagggtc 8100
ggaggaggca agagacatgg gcggtactgg attgccgaca gggcaatcaa ctcgtcctcc 8160
actcccactc tgtctcaggt gagagcaagg agcatgggct cgagcccagc cattcgacct 8220
cggcacgaca gctcacatca tcgcatacag caactcgagg ttagtacttc tataactcgt 8280
ccttccttta gttatatatc tagtctttga gttactataa cgttggcttg taatattaca 8340
gacccaacta gaagagatgg aggcgaggat aatggcggag cgggcggcgg ctgatcagag 8400
gatggcggag atgttccagt acatgcagag ccttggcgcc gcacagggca tcgctccgcc 8460
acctccattg ttccccccag ttgaccctac tctcttccac actcctgtga gtatcaaaat 8520
tgtagtttga tgtttgtaat gcatctggta taacacatgc tatctcttct ctgtacgaat 8580
caagctgatt catcagtata ggnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 8640
nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnnnnnnnnn 8700
nnatctgtgg gttgaacatc aaaataaaaa atcaaatgac caatacagca tagtatgcta 8760
cgtgctccag ggatggggat caacttcggt gatgcgaggg ggaactgggt tctataagaa 8820
gtggtcgaga tatgaagcat ctgatcgagg tctcatcacc atgagaactg tgttgactca 8880
tgccattatc aacgctattc aacttgagta ttagtagatg ctgcaggccc ttgcgtaccg 8940
gttggagtaa atatgacctt agtctgatta tgcacctcca gagacaagat aagcaagcaa 9000
atcccgcacc ctaacccgac catcagactc aaccaatgga agacacccct cctcgacaac 9060
atctgccccg agtgtcccaa atggcactcg gcaaagaagg cggctttgcc gagtgctgcc 9120
aggaggacac tcggcaaaga tatcttcgtt gccgagtgtc aacggtgaca ctcggcaaag 9180
ccgccgtctc cgtcaaccgg cgccgtaacg gtcgcttttc tttgccgagt gctccctgac 9240
actcggcaaa gatcttcgcc gagtgcccga gaaaaagtac tcggcaaaga agtctttgcc 9300
gatgcactgt ttgccgagcc ttctttgccg agtgttacac tcggcaaagc ctttgccgag 9360
tgtttttagg gcttcgccga gtgcttccgg cactcggcga agctattgat tccggtagtg 9420
ttggtagcta ggcaagctaa tggggtacat atggaaggag gaaaccaagt cgatcgtcgt 9480
cgtagcatgt cggtgtgggt actacactac acacacatat acatgggcaa cgcaaggcca 9540
cctttctgaa tcctgcatga gcgtgtacca ctagaattgt cagtgtgtgc ggtgtatggc 9600
aggtttttgg ttcggcaagt ggggccctcc ggggaggaat ctcagtaaca aaccgctctt 9660
ctgaaaaggt cagccatccc cggtccggtc cggtgatgtc gtcgctgtcg ctctgctagc 9720
ttgctgccga tccccccccc cccccccccc cttcttctct ctacccctcc ctccacctca 9780
taaatactta gtttaataac cttgcactgc cgcagtagcc cttaactgct gctatctatc 9840
tcttttctga aggaaaaaaa aggtttgata ctcctctacc tagctagtcc tgcatgccgc 9900
taatgtgcgt cttgcctgtt tatttgttct taataagggc tgcctatcta ttatattttg 9960
cacctgtttt gctgtgttct tggtaactag cttaattcct tcgcctacaa tcgtcaaatc 10020
ccccccatca tcagtcagat gaacttttga tcgaattgaa gttgttcttc taattcggcc 10080
ccagcagcgc ccatgcatct ggttttattt gctttctgtt gggtataata tgcaagacct 10140
tttgttgcta gggcaaggct gcaaccacat gcgtgtactg aactcatgat gtaactcatc 10200
ctttttgttt gctcacagaa tcactactct actgcacttc cttttcatcc gatccgcaat 10260
cttttttttc ttttacatgc tttagttttc tctctttctt gattacaaac atgattactg 10320
gaactttctt aggctgcctt ccccttcctt ggatctgctt tagttttctt ttttgggcta 10380
ccgcgcgcgg cttatttgag tttatcactt gctgcatata cataatatat atatacatgc 10440
atgcgatggc gttcatgtta ctcaactaca gatctgtttc tgttcgtgtg tttcagttca 10500
gcgcgcagtt aagcatagca ggacgaccac gacgatgtat cacccgcagt gcgagctcct 10560
gacgatggcg cacgaaacgc cggacctgga cgccggccag ccgcacctaa ccgtctccgg 10620
cgtcgccagc atcccggcag agctgagctt ccacctgctg cactcgctcg acgccgcggc 10680
ggcggtcaat cccgtcacgg cgccgccgca gtccaccatc gactacttcc tcggcggcgc 10740
cgatccccac cagcaggcca tgcagtacga gccgctgccg cccgccgcgg gcggccacca 10800
ccagtacacc atggacatgt tccgcgacta ctgcgacggc cactacccca ccgccgagcc 10860
gtacatccgc gggacaatga ctggagccct cgtgttcggg gccaccgacg acgacgactc 10920
ggccgctgcc tacatgcccg gggggcactt tgagacctcc ccgccgccgc cacgcgccac 10980
cggccgcggc aggaagcggg gcagggcgct gggcggcggc ttccatgctg tgctggccaa 11040
cggcgtcgag aagaaggaga agcagcgccg gctgcggctc accgagaagt acaccgccct 11100
catgcacctc atacccaacg ttacaaaggt cgtaccaaat cctcctctta tgttcgtcca 11160
tcgtttcaaa ttaagttaaa aaattaattc acggttcttg ttgtttattt tttgcgcact 11220
gcagactgat agggcgacgg tgatctcgga cgcgatcgag tacatccagg agctggggag 11280
gacggtggag gagctgacgc tgctggtgga gaagaagcgg cgccggaggg agctgcaggg 11340
ggacgtcgtg gacgcggcgc cggctgcggt ggttgctgcc gccggtgagg cggagagctc 11400
ggagggcgag gtggctcctc cgccgccggc cgtgccgcgg cagccgatcc ggagcacgta 11460
catccagcgg cggagcaagg acacgtccgt ggacgtgcgg atcgtggagg aggacgtgaa 11520
catcaagctc accaagcgcc ggcgcgacgg gtgcctcgca gccgcgtcgc gcgcgctgga 11580
tgacctccgc cttgacctcg tccacctctc cggcggcaag atcggtgact gtcaaatcta 11640
catgttcaac accaaggtac atacgaatac gatacgtagc cattgatcga tctgtaattc 11700
tgtagcctga cgattccgag gtttctggtg ctaaaaaatg catctttttt tctcagatga 11760
caatgctttc tgtctttgtt caccgcagat tcacaagggg tcttcagtgt ttgcgagtgc 11820
agtggccggt aggctgatgg aagtggtgga cgagtactag gctaccatgc acttgaattt 11880
ctagctagct ctacgtaccg cgctgctatg aatctagcta tagcgtttct tggatgaaag 11940
ac 11942
<210> 218
<211> 3197
<212> ДНК
<213> Zea mays
<400> 218
ggcaagctaa tggggtacat atggaaggag gaaaccaagt cgatcgtcgt cgtagcatgt 60
cggtgtgggt actacactac acacacatat acatgggcaa cgcaaggcca cctttctgaa 120
tcctgcatga gcgtgtacca ctagaattgt cagtgtgtgc ggtgtatggc aggtttttgg 180
ttcggcaagt ggggccctcc ggggaggaat ctcagtaaca aaccgctctt ctgaaaaggt 240
cagccatccc cggtccggtc cggtgatgtc gtcgctgtcg ctctgctagc ttgctgccga 300
tccccccccc cccccccccc cttcttctct ctacccctcc ctccacctca taaatactta 360
gtttaataac cttgcactgc cgcagtagcc cttaactgct gctatctatc tcttttctga 420
aggaaaaaaa aggtttgata ctcctctacc tagctagtcc tgcatgccgc taatgtgcgt 480
cttgcctgtt tatttgttct taataagggc tgcctatcta ttatattttg cacctgtttt 540
gctgtgttct tggtaactag cttaattcct tcgcctacaa tcgtcaaatc ccccccatca 600
tcagtcagat gaacttttga tcgaattgaa gttgttcttc taattcggcc ccagcagcgc 660
ccatgcatct ggttttattt gctttctgtt gggtataata tgcaagacct tttgttgcta 720
gggcaaggct gcaaccacat gcgtgtactg aactcatgat gtaactcatc ctttttgttt 780
gctcacagaa tcactactct actgcacttc cttttcatcc gatccgcaat cttttttttc 840
ttttacatgc tttagttttc tctctttctt gattacaaac atgattactg gaactttctt 900
aggctgcctt ccccttcctt ggatctgctt tagttttctt ttttgggcta ccgcgcgcgg 960
cttatttgag tttatcactt gctgcatata cataatatat atatacatgc atgcgatggc 1020
gttcatgtta ctcaactaca gatctgtttc tgttcgtgtg tttcagttca gcgcgcagtt 1080
aagcatagca ggacgaccac gacgatgtat cacccgcagt gcgagctcct gacgatggcg 1140
cacgaaacgc cggacctgga cgccggccag ccgcacctaa ccgtctccgg cgtcgccagc 1200
atcccggcag agctgagctt ccacctgctg cactcgctcg acgccgcggc ggcggtcaat 1260
cccgtcacgg cgccgccgca gtccaccatc gactacttcc tcggcggcgc cgatccccac 1320
cagcaggcca tgcagtacga gccgctgccg cccgccgcgg gcggccacca ccagtacacc 1380
atggacatgt tccgcgacta ctgcgacggc cactacccca ccgccgagcc gtacatccgc 1440
gggacaatga ctggagccct cgtgttcggg gccaccgacg acgacgactc ggccgctgcc 1500
tacatgcccg gggggcactt tgagacctcc ccgccgccgc cacgcgccac cggccgcggc 1560
aggaagcggg gcagggcgct gggcggcggc ttccatgctg tgctggccaa cggcgtcgag 1620
aagaaggaga agcagcgccg gctgcggctc accgagaagt acaccgccct catgcacctc 1680
atacccaacg ttacaaaggt cgtaccaaat cctcctctta tgttcgtcca tcgtttcaaa 1740
ttaagttaaa aaattaattc acggttcttg ttgtttattt tttgcgcact gcagactgat 1800
agggcgacgg tgatctcgga cgcgatcgag tacatccagg agctggggag gacggtggag 1860
gagctgacgc tgctggtgga gaagaagcgg cgccggaggg agctgcaggg ggacgtcgtg 1920
gacgcggcgc cggctgcggt ggttgctgcc gccggtgagg cggagagctc ggagggcgag 1980
gtggctcctc cgccgccggc cgtgccgcgg cagccgatcc ggagcacgta catccagcgg 2040
cggagcaagg acacgtccgt ggacgtgcgg atcgtggagg aggacgtgaa catcaagctc 2100
accaagcgcc ggcgcgacgg gtgcctcgca gccgcgtcgc gcgcgctgga tgacctccgc 2160
cttgacctcg tccacctctc cggcggcaag atcggtgact gtcaaatcta catgttcaac 2220
accaaggtac atacgaatac gatacgtagc cattgatcga tctgtaattc tgtagcctga 2280
cgattccgag gtttctggtg ctaaaaaatg catctttttt tctcagatga caatgctttc 2340
tgtctttgtt caccgcagat tcacaagggg tcttcagtgt ttgcgagtgc agtggccggt 2400
aggctgatgg aagtggtgga cgagtactag gctaccatgc acttgaattt ctagctagct 2460
ctacgtaccg cgctgctatg aatctagcta tagcgtttct tggatgaaag actagttagt 2520
tgttaccttc tatctttgct tcaattaaat ccgcttgctc gttacagact gagtttgttt 2580
ctaaatgtca aggttgtttt ggtcaaattg aataaattgg cacactggcc tgtgaggtta 2640
ttatatatat ttatgtgttt attactggtc tattaatttg tcttattatt aatgtattgc 2700
ctgtcaagga ataaatggta tgatgaccat atttatgcat agataggatc ggatgagtag 2760
gttcacttgc ttgagttcac cggtataatt ccggatacat ctggttaggt catcctttgg 2820
tcagctgccc gcaagcttaa ctccgtgcga tatacaatat acagatttta ttatggtttt 2880
cccctgaacc ttcgtgacta actatgttat catttttata gctttatagt ctacaaactg 2940
ttttatactc agcttgataa gtacattctg gtttggacga tggttttttt ttcttgcaaa 3000
atgaatttgt cttcagcctt tacgactaca tacagtttag tttgtattaa ttgataccgg 3060
aagatcagat tcggaccaca tataaacaag gaatatatag cacgtactcg ctgaacctta 3120
aatatagtca ggaaaataga gggttaacta aaccgatcca gaaaccaatt acattgatat 3180
tgactctatt cttcgtt 3197
<210> 219
<211> 3173
<212> ДНК
<213> Zea mays
<400> 219
ggcaagctaa tggggtacat atggaaggag gaaaccaagt cgatcgtcgt cgtagcatgt 60
cggtgtgggt actacactac acacacatat acatgggcaa cgcaaggcca cctttctgaa 120
tcctgcatga gcgtgtacca ctagaattgt cagtgtgtgc ggtgtatggc aggtttttgg 180
ttcggcaagt ggggccctcc ggggaggaat ctcagtaaca aaccgctctt ctgaaaaggt 240
cagccatccc cggtccggtc cggtgatgtc gtcgctgtcg ctctgctagc ttgctgccga 300
tccccccccc ttcttctctc tacccctccc tccacctcat aaatacttag tttaataacc 360
ttgcactgcc gcagtagccc ttaactgctg ctatctatct cttttctgaa ggaaaaaaaa 420
ggtttgatac tcctctacct agctagtcct gcatgccgct aatgtgcgtc ttgcctgttt 480
atttgttctt aataagggct gcctatctat tatattttgc acctgttttg ctgtgttctt 540
ggtaactagc ttaattcctt tgcctacaat cgtcaaatcc cccccatcat cagtcagatg 600
aacttttgat cgaattgaag ttgttcttct aattcggccc cagcagcgcc catgcatctg 660
gttttatttg ctttctgttg ggtataatat gcaagacctt ttgttgctag ggcaaggctg 720
caaccacatg cgtgtactga actcatgatg taactcatcc tttttgtttg ctcacagaat 780
cactactcta ctgcacttcc ttttcatccg atccgcaatc ttttttttct tttacatgct 840
ttagttttct ctctttcttg attacaaaca tgattactgg aactttctta ggctgccttc 900
cccttccttg gatctgcttt agttttcttt tttgggctac cgcgcgcggc ttatttgagt 960
ttatcacttg ctgcatatac ataatatata tatacatgca tgcgatggcg ttcatgttac 1020
tcaactacag atctgtttct gttcgtgtgt ttcagttcag cgcgcagtta agcatagcag 1080
gacgaccacg acgatgtatc acccgcagtg cgagctcctg acgatggcgc acgaaacgcc 1140
ggacctggac gccggccagc cgcacctaac cgtctccggc gtcgccagca tcccggcaga 1200
gctgagcttc cacctgctgc actcgctcga cgccgcggcg gcggtcaatc ccgtcacggc 1260
gccgccgcag tccaccatcg actacttcct cggcggcgcc gatccccacc agcaggccat 1320
gcagtacgag ccgctgccgc ccgccgcggg cggccaccac cagtacacca tggacatgtt 1380
ccgcgactac tgcgacggcc actaccccac cgccgagccg tacatccgcg ggacaatgac 1440
tggagccctc gtgttcgggg ccaccgacga cgacgactcg gccgctgcct acatgcccgg 1500
ggggcacttt gagacctccc cgccgccgcc acgcgccacc ggccgcggca ggaagcgggg 1560
cagggcgctg ggcggcggct tccatgctgt gctggccaac ggcgtcgaga agaaggagaa 1620
gcagcgccgg ctgcggctca ccgagaagta caccgccctc atgcacctca tacccaacgt 1680
tacaaaggtc gtaccaaatc ctcctcttat gttcgtccat cgtttgaaat taagttaaaa 1740
aattaattca cggttcttgt tgtttatttt ttgcgcactg cagactgata gggcgacggt 1800
gatctcggac gcgatcgagt acatccagga gctggggagg acggtggagg agctgacgct 1860
gctggtggag aagaagcggc gccggaggga gctgcagggg gacgtcgtgg acgcggcgcc 1920
ggctgcggtg gttgctgccg ccggtgaggc ggagagctcg gagggcgagg tggctcctcc 1980
gccgccggcc gtgccgcggc agccgatccg gagcacgtac atccagcggc ggagcaagga 2040
cacgtccgtg gacgtgcgga tcgtggagga ggacgtgaac atcaagctca ccaagcgccg 2100
gcgcgacggg tgcctcgcag ccgcgtcgcg cgcgctggat gacctccgcc ttgacctcgt 2160
ccacctctcc ggcggcaaga tcggtgactg tcaaatctac atgttcaaca ccaaggtaca 2220
tacgaatacg atacgtagcc attgatcgat ctgtaattct gtagcctgac gattccgagg 2280
tttctggtgc taaaaaatgc atcttttttt ctcagatgac aatgctttct gtctttgttc 2340
accgcagatt cacaaggggt cttcagtgtt tgcgagtgca gtggccggta ggctgatgga 2400
agtggtggac gagtactagg ctaccatgca cttgaatttc tagctagctc tacgtaccgc 2460
gctgctatga atctagctat agcgtttctt ggatgaaaga ctagttagtt gttaccttct 2520
atctttgctt caattaaatc cgcttgctcg ttacagactg agtttgtttc taaatgtcaa 2580
ggttgttttg gtcaaattga ataaattggc acactggcct gtgaggttat tatatatatt 2640
tatgtgttta ttactggtct attaatttgt cctattatta atgtattgcc tgtcaaggaa 2700
taaatgatat gatgaccata tttatgcata gataggatga gtaggttcac ttgcttgagt 2760
tcaccggtat aattctggat acatctggtt aggtcatcct ttggtcagct gcccgcaacg 2820
tgcgatatac aatatacata ttttattatg tttttttcgt gactaactat gttatcattt 2880
ttatagcttt atagtctaca aactgtttta tactcagctt gataagtaca ttctggtttg 2940
gacgattttt ttttcttgca aaaatgaatt tgtcttcagc ctttacgact acatacagtt 3000
tagttcttag agtatctcat ctgtattaat tgataccgga agagattcgg gccacatata 3060
aacaaggaat atatagcacg tactcgctga accttaaata tagtcaggaa catagagggt 3120
taactaaacc gatccagaaa ccaattacat tgatattgac tctattcttc gtt 3173
<210> 220
<211> 3235
<212> ДНК
<213> Zea mays
<400> 220
ggcaagctaa tggggtacat atggaaggag gaaaccaagt cgatcgtcgt cgtagcatgt 60
cggtgtgggt actacactac acacacatat acatgggcaa cgcaaggcca cctttctgaa 120
tcctgcatga gcgtgtacca ctagaattgt cagtgtgtgc ggtgtatggc aggtttttgg 180
ttcggcaagt ggggccctcc ggggaggaat ctcagtaaca aaccgctctt ctgaaaaggt 240
cagccatccc cggtccggtc cggtgatgtc gtcgctgtcg ctctgctagc ttgctgccga 300
tccccccccc cccccccccc cttcttctct ctacccctcc ctccacctca taaatactta 360
gtttaataac cttgcactgc cgcagtagcc cttaactgct gctatctatc tcttttctga 420
aggaaaaaaa aggtttgata ctcctctacc tagctagtcc tgcatgccgc taatgtgcgt 480
cttgcctgtt tatttgttct taataagggc tgcctatcta ttatattttg cacctgtttt 540
gctgtgttct tggtaactag cttaattcct ttgcctacaa tcgtcaaatc ccccccatca 600
tcagtcagat gaacttttga tcgaattgaa gttgttcttc taattcggcc ccagcagcgc 660
ccatgcatct ggttttattt gctttctgtt gggtataata tgcaagacct tttgttgcta 720
gggcaaggct gcaaccacat gcgtgtactg aactcatgat gtaactcatc ctttttgttt 780
gctcacagaa tcactactct actgcacttc cttttcatcc gatccgcaat cttttttttc 840
ttttacatgc tttagttttc tctctttctt gattacaaac atgattactg gaactttctt 900
aggctgcctt ccccttcctt ggatctgctt tagttttctt ttttgggcta ccgcgcgcgg 960
cttatttgag tttatcactt gctgcatata cataatatat atatacatgc atgcgatggc 1020
gttcatgtta ctcaactaca gatctgtttc tgttcgtgtg tttcagttca gcgcgcagtt 1080
aagcatagca ggacgaccac gacgatgtat cacccgcagt gcgagctcct gacgatggcg 1140
cacgaaacgc cggacctgga cgccggccag ccgcacctaa ccgtctccgg cgtcgccagc 1200
atcccggcag agctgagctt ccacctgctg cactcgctcg acgccgcggc ggcggtcaat 1260
cccgtcacgg cgccgccgca gtccaccatc gactacttcc tcggcggcgc cgatccccac 1320
cagcaggcca tgcagtacga gccgctgccg cccgccgcgg gcggccacca ccagtacacc 1380
atggacatgt tccgcgacta ctgcgacggc aactacccca ccgccgagcc gtacatccgc 1440
gggacaatga ctggagccct cgtgttcggg gccaccgacg acgacgactc ggccgctgcc 1500
gcctacatgc ccggggggca ctttgagacc tccccgccgc cgccacgcgc caccggccgc 1560
ggcaggaagc ggggcagggc gctgggcggc ggcttccatg ctgtgctggc caacggcgtc 1620
gagaagaagg agaagcagcg ccggctgcgg ctcaccgaga agtttacggc cctcatgcac 1680
ctcataccca acgttacgaa ggtcgtacgg cgtacttgcg cgcggaccaa atcctcctct 1740
tatgttcgtc gtccatcgtc tcaaattaat tcacggttct tgttgttgtt tattttttgc 1800
gcactgcaga ctgatagggc gacggtgatc tcggacgcga tcgagtacat ccaggagctg 1860
gggaggacgg tggaggagct gacgctgctg gtggagaaga agcggcgccg gagggagctg 1920
cagggggacg tcgtggacgc ggcgccggct gcggtggttg ctgccgccgg tgaggcggag 1980
agctcggagg gcgaggtggc tcctccgccg ctggccgtgc cgcggcagcc gatccggagc 2040
acgtacatcc agcggcggag caaggacacg tccgtggacg tgcggatcgt ggaggaggac 2100
gtgaacatca agctcaccaa gcgccggcgc gacgggtgcc tcgcagccgc gtcgcgcgcg 2160
ctggacgacc tccgccttga cctcgtccac ctctccggcg gcaagatcgg tgactgtcaa 2220
atctacatgt tcaacaccaa ggtacatacg aatacgatac gtagccattg atcgatctgt 2280
aattctgtag cctgacgatt tcatgcatta cttttccgag gtttctgtgc tatactacct 2340
aacctaggtg ctaaaaaatg cacctttttt tctcagatga caatgctttc tgtctttgtt 2400
caccgcagat tcacaagggg tcttcagtgt ttgcgagtgc agtggccggt aggctgatgg 2460
aagtggtgga cgagtactag gctaccatgc acttgaattt ctagctagct ctacgtaccg 2520
cgctgctatg aatctagcta tagcgtttct tggatgaaag aatagttagt tgttaccttc 2580
tatctttgct tcaattaaat ccgcttgctc gttacagact gagtttgttt ctaaatgtca 2640
aggttgtttt ggtcaaattg aataaattgg cacactggcc tgtgaggtta ttatatttat 2700
gtgtattatt actggtctat caatttgtcc tattattgta ttgcctgtca aggaataaat 2760
tgtatgatga tcatatttat gcatagatag gatgagtagg ttcacttgct tgagttcacc 2820
ggtataattc tggtttctgg atacatctgg ttaggtcagc ctttggtcag ctgcccgcaa 2880
gcttaactcc gtgcgatata cactatacaa attttattat gtttttttcg tgactaacta 2940
tgttatcatt tttatagctt tatagtctac aaactgtttt atactcagct tgataagtac 3000
attctggttt ggacgatggt tttttttttc ttgcaaaaat gaatttgtct tcagccttta 3060
cgactacata cagtttagtt tgtattaatt gataccagaa gatcagattc ggaccacata 3120
taaacaagga atatatagca cgtactcgct gaaccttaaa tatagtcagg aacatagagg 3180
gttaactaaa ccgatccaga aaccaattac attgatattg actctattct tcgtt 3235
<210> 221
<211> 1495
<212> ДНК
<213> Zea mays
<400> 221
ctctgctagc ttgctgccga tccccccccc cccccccccc cttcttctct ctacccctcc 60
ctccacctca taaatactta gtttaataac cttgcactgc cgcagtagcc cttaactgct 120
gctatctatc tcttttctga aggaaaaaaa agttcagcgc gcagttaagc atagcaggac 180
gaccacgacg atgtatcacc cgcagtgcga gctcctgacg atggcgcacg aaacgccgga 240
cctggacgcc ggccagccgc acctaaccgt ctccggcgtc gccagcatcc cggcagagct 300
gagcttccac ctgctgcact cgctcgacgc cgcggcggcg gtcaatcccg tcacggcgcc 360
gccgcagtcc accatcgact acttcctcgg cggcgccgat ccccaccagc aggccatgca 420
gtacgagccg ctgccgcccg ccgcgggcgg ccaccaccag tacaccatgg acatgttccg 480
cgactactgc gacggccact accccaccgc cgagccgtac atccgcggga caatgactgg 540
agccctcgtg ttcggggcca ccgacgacga cgactcggcc gctgcctaca tgcccggggg 600
gcactttgag acctccccgc cgccgccacg cgccaccggc cgcggcagga agcggggcag 660
ggcgctgggc ggcggcttcc atgctgtgct ggccaacggc gtcgagaaga aggagaagca 720
gcgccggctg cggctcaccg agaagtacac cgccctcatg cacctcatac ccaacgttac 780
aaagactgat agggcgacgg tgatctcgga cgcgatcgag tacatccagg agctggggag 840
gacggtggag gagctgacgc tgctggtgga gaagaagcgg cgccggaggg agctgcaggg 900
ggacgtcgtg gacgcggcgc cggctgcggt ggttgctgcc gccggtgagg cggagagctc 960
ggagggcgag gtggctcctc cgccgccggc cgtgccgcgg cagccgatcc ggagcacgta 1020
catccagcgg cggagcaagg acacgtccgt ggacgtgcgg atcgtggagg aggacgtgaa 1080
catcaagctc accaagcgcc ggcgcgacgg gtgcctcgca gccgcgtcgc gcgcgctgga 1140
tgacctccgc cttgacctcg tccacctctc cggcggcaag atcggtgact gtcaaatcta 1200
catgttcaac accaagattc acaaggggtc ttcagtgttt gcgagtgcag tggccggtag 1260
gctgatggaa gtggtggacg agtactaggc taccatgcac ttgaatttct agctagctct 1320
acgtaccgcg ctgctatgaa tctagctata gcgtttcttg gatgaaagac tagttagttg 1380
ttaccttcta tctttgcttc aattaaatcc gcttgctcgt tacagactga gtttgtttct 1440
aaatgtcaag gttgttttgg tcaaattgaa taaattggca cactggcctg tgagg 1495
<210> 222
<211> 1484
<212> ДНК
<213> Zea mays
<400> 222
ctctgctagc ttgctgccga tccccccccc ttcttctctc tacccctccc tccacctcat 60
aaatacttag tttaataacc ttgcactgcc gcagtagccc ttaactgctg ctatctatct 120
cttttctgaa ggaaaaaaaa gttcagcgcg cagttaagca tagcaggacg accacgacga 180
tgtatcaccc gcagtgcgag ctcctgacga tggcgcacga aacgccggac ctggacgccg 240
gccagccgca cctaaccgtc tccggcgtcg ccagcatccc ggcagagctg agcttccacc 300
tgctgcactc gctcgacgcc gcggcggcgg tcaatcccgt cacggcgccg ccgcagtcca 360
ccatcgacta cttcctcggc ggcgccgatc cccaccagca ggccatgcag tacgagccgc 420
tgccgcccgc cgcgggcggc caccaccagt acaccatgga catgttccgc gactactgcg 480
acggccacta ccccaccgcc gagccgtaca tccgcgggac aatgactgga gccctcgtgt 540
tcggggccac cgacgacgac gactcggccg ctgcctacat gcccgggggg cactttgaga 600
cctccccgcc gccgccacgc gccaccggcc gcggcaggaa gcggggcagg gcgctgggcg 660
gcggcttcca tgctgtgctg gccaacggcg tcgagaagaa ggagaagcag cgccggctgc 720
ggctcaccga gaagtacacc gccctcatgc acctcatacc caacgttaca aagactgata 780
gggcgacggt gatctcggac gcgatcgagt acatccagga gctggggagg acggtggagg 840
agctgacgct gctggtggag aagaagcggc gccggaggga gctgcagggg gacgtcgtgg 900
acgcggcgcc ggctgcggtg gttgctgccg ccggtgaggc ggagagctcg gagggcgagg 960
tggctcctcc gccgccggcc gtgccgcggc agccgatccg gagcacgtac atccagcggc 1020
ggagcaagga cacgtccgtg gacgtgcgga tcgtggagga ggacgtgaac atcaagctca 1080
ccaagcgccg gcgcgacggg tgcctcgcag ccgcgtcgcg cgcgctggat gacctccgcc 1140
ttgacctcgt ccacctctcc ggcggcaaga tcggtgactg tcaaatctac atgttcaaca 1200
ccaagattca caaggggtct tcagtgtttg cgagtgcagt ggccggtagg ctgatggaag 1260
tggtggacga gtactaggct accatgcact tgaatttcta gctagctcta cgtaccgcgc 1320
tgctatgaat ctagctatag cgtttcttgg atgaaagact agttagttgt taccttctat 1380
ctttgcttca attaaatccg cttgctcgtt acagactgag tttgtttcta aatgtcaagg 1440
ttgttttggt caaattgaat aaattggcac actggcctgt gagg 1484
<210> 223
<211> 1498
<212> ДНК
<213> Zea mays
<400> 223
ctctgctagc ttgctgccga tccccccccc cccccccccc cttcttctct ctacccctcc 60
ctccacctca taaatactta gtttaataac cttgcactgc cgcagtagcc cttaactgct 120
gctatctatc tcttttctga aggaaaaaaa agttcagcgc gcagttaagc atagcaggac 180
gaccacgacg atgtatcacc cgcagtgcga gctcctgacg atggcgcacg aaacgccgga 240
cctggacgcc ggccagccgc acctaaccgt ctccggcgtc gccagcatcc cggcagagct 300
gagcttccac ctgctgcact cgctcgacgc cgcggcggcg gtcaatcccg tcacggcgcc 360
gccgcagtcc accatcgact acttcctcgg cggcgccgat ccccaccagc aggccatgca 420
gtacgagccg ctgccgcccg ccgcgggcgg ccaccaccag tacaccatgg acatgttccg 480
cgactactgc gacggcaact accccaccgc cgagccgtac atccgcggga caatgactgg 540
agccctcgtg ttcggggcca ccgacgacga cgactcggcc gctgccgcct acatgcccgg 600
ggggcacttt gagacctccc cgccgccgcc acgcgccacc ggccgcggca ggaagcgggg 660
cagggcgctg ggcggcggct tccatgctgt gctggccaac ggcgtcgaga agaaggagaa 720
gcagcgccgg ctgcggctca ccgagaagtt tacggccctc atgcacctca tacccaacgt 780
tacgaagact gatagggcga cggtgatctc ggacgcgatc gagtacatcc aggagctggg 840
gaggacggtg gaggagctga cgctgctggt ggagaagaag cggcgccgga gggagctgca 900
gggggacgtc gtggacgcgg cgccggctgc ggtggttgct gccgccggtg aggcggagag 960
ctcggagggc gaggtggctc ctccgccgct ggccgtgccg cggcagccga tccggagcac 1020
gtacatccag cggcggagca aggacacgtc cgtggacgtg cggatcgtgg aggaggacgt 1080
gaacatcaag ctcaccaagc gccggcgcga cgggtgcctc gcagccgcgt cgcgcgcgct 1140
ggacgacctc cgccttgacc tcgtccacct ctccggcggc aagatcggtg actgtcaaat 1200
ctacatgttc aacaccaaga ttcacaaggg gtcttcagtg tttgcgagtg cagtggccgg 1260
taggctgatg gaagtggtgg acgagtacta ggctaccatg cacttgaatt tctagctagc 1320
tctacgtacc gcgctgctat gaatctagct atagcgtttc ttggatgaaa gaatagttag 1380
ttgttacctt ctatctttgc ttcaattaaa tccgcttgct cgttacagac tgagtttgtt 1440
tctaaatgtc aaggttgttt tggtcaaatt gaataaattg gcacactggc ctgtgagg 1498
<210> 224
<211> 365
<212> БЕЛОК
<213> Zea mays
<400> 224
Met Tyr His Pro Gln Cys Glu Leu Leu Thr Met Ala His Glu Thr Pro
1 5 10 15
Asp Leu Asp Ala Gly Gln Pro His Leu Thr Val Ser Gly Val Ala Ser
20 25 30
Ile Pro Ala Glu Leu Ser Phe His Leu Leu His Ser Leu Asp Ala Ala
35 40 45
Ala Ala Val Asn Pro Val Thr Ala Pro Pro Gln Ser Thr Ile Asp Tyr
50 55 60
Phe Leu Gly Gly Ala Asp Pro His Gln Gln Ala Met Gln Tyr Glu Pro
65 70 75 80
Leu Pro Pro Ala Ala Gly Gly His His Gln Tyr Thr Met Asp Met Phe
85 90 95
Arg Asp Tyr Cys Asp Gly His Tyr Pro Thr Ala Glu Pro Tyr Ile Arg
100 105 110
Gly Thr Met Thr Gly Ala Leu Val Phe Gly Ala Thr Asp Asp Asp Asp
115 120 125
Ser Ala Ala Ala Tyr Met Pro Gly Gly His Phe Glu Thr Ser Pro Pro
130 135 140
Pro Pro Arg Ala Thr Gly Arg Gly Arg Lys Arg Gly Arg Ala Leu Gly
145 150 155 160
Gly Gly Phe His Ala Val Leu Ala Asn Gly Val Glu Lys Lys Glu Lys
165 170 175
Gln Arg Arg Leu Arg Leu Thr Glu Lys Tyr Thr Ala Leu Met His Leu
180 185 190
Ile Pro Asn Val Thr Lys Thr Asp Arg Ala Thr Val Ile Ser Asp Ala
195 200 205
Ile Glu Tyr Ile Gln Glu Leu Gly Arg Thr Val Glu Glu Leu Thr Leu
210 215 220
Leu Val Glu Lys Lys Arg Arg Arg Arg Glu Leu Gln Gly Asp Val Val
225 230 235 240
Asp Ala Ala Pro Ala Ala Val Val Ala Ala Ala Gly Glu Ala Glu Ser
245 250 255
Ser Glu Gly Glu Val Ala Pro Pro Pro Pro Ala Val Pro Arg Gln Pro
260 265 270
Ile Arg Ser Thr Tyr Ile Gln Arg Arg Ser Lys Asp Thr Ser Val Asp
275 280 285
Val Arg Ile Val Glu Glu Asp Val Asn Ile Lys Leu Thr Lys Arg Arg
290 295 300
Arg Asp Gly Cys Leu Ala Ala Ala Ser Arg Ala Leu Asp Asp Leu Arg
305 310 315 320
Leu Asp Leu Val His Leu Ser Gly Gly Lys Ile Gly Asp Cys Gln Ile
325 330 335
Tyr Met Phe Asn Thr Lys Ile His Lys Gly Ser Ser Val Phe Ala Ser
340 345 350
Ala Val Ala Gly Arg Leu Met Glu Val Val Asp Glu Tyr
355 360 365
<210> 225
<211> 366
<212> БЕЛОК
<213> Zea mays
<400> 225
Met Tyr His Pro Gln Cys Glu Leu Leu Thr Met Ala His Glu Thr Pro
1 5 10 15
Asp Leu Asp Ala Gly Gln Pro His Leu Thr Val Ser Gly Val Ala Ser
20 25 30
Ile Pro Ala Glu Leu Ser Phe His Leu Leu His Ser Leu Asp Ala Ala
35 40 45
Ala Ala Val Asn Pro Val Thr Ala Pro Pro Gln Ser Thr Ile Asp Tyr
50 55 60
Phe Leu Gly Gly Ala Asp Pro His Gln Gln Ala Met Gln Tyr Glu Pro
65 70 75 80
Leu Pro Pro Ala Ala Gly Gly His His Gln Tyr Thr Met Asp Met Phe
85 90 95
Arg Asp Tyr Cys Asp Gly Asn Tyr Pro Thr Ala Glu Pro Tyr Ile Arg
100 105 110
Gly Thr Met Thr Gly Ala Leu Val Phe Gly Ala Thr Asp Asp Asp Asp
115 120 125
Ser Ala Ala Ala Ala Tyr Met Pro Gly Gly His Phe Glu Thr Ser Pro
130 135 140
Pro Pro Pro Arg Ala Thr Gly Arg Gly Arg Lys Arg Gly Arg Ala Leu
145 150 155 160
Gly Gly Gly Phe His Ala Val Leu Ala Asn Gly Val Glu Lys Lys Glu
165 170 175
Lys Gln Arg Arg Leu Arg Leu Thr Glu Lys Phe Thr Ala Leu Met His
180 185 190
Leu Ile Pro Asn Val Thr Lys Thr Asp Arg Ala Thr Val Ile Ser Asp
195 200 205
Ala Ile Glu Tyr Ile Gln Glu Leu Gly Arg Thr Val Glu Glu Leu Thr
210 215 220
Leu Leu Val Glu Lys Lys Arg Arg Arg Arg Glu Leu Gln Gly Asp Val
225 230 235 240
Val Asp Ala Ala Pro Ala Ala Val Val Ala Ala Ala Gly Glu Ala Glu
245 250 255
Ser Ser Glu Gly Glu Val Ala Pro Pro Pro Leu Ala Val Pro Arg Gln
260 265 270
Pro Ile Arg Ser Thr Tyr Ile Gln Arg Arg Ser Lys Asp Thr Ser Val
275 280 285
Asp Val Arg Ile Val Glu Glu Asp Val Asn Ile Lys Leu Thr Lys Arg
290 295 300
Arg Arg Asp Gly Cys Leu Ala Ala Ala Ser Arg Ala Leu Asp Asp Leu
305 310 315 320
Arg Leu Asp Leu Val His Leu Ser Gly Gly Lys Ile Gly Asp Cys Gln
325 330 335
Ile Tyr Met Phe Asn Thr Lys Ile His Lys Gly Ser Ser Val Phe Ala
340 345 350
Ser Ala Val Ala Gly Arg Leu Met Glu Val Val Asp Glu Tyr
355 360 365
<210> 226
<211> 366
<212> БЕЛОК
<213> Brachypodium distachyon
<400> 226
Met Tyr His Pro Gln Cys Glu Leu Leu Met Pro His Glu Ser Leu Asp
1 5 10 15
Met Asp Ala Val Val Gly Gln Ser His Leu Ala Ala Ser Gly Val Ser
20 25 30
Ala Ile Pro Ala Glu Leu Asn Phe His Leu Leu His His Ser Phe Val
35 40 45
Asp Thr Ala Ala Ser Pro Gln Pro Pro Thr Val Asp Tyr Phe Phe Pro
50 55 60
Gly Thr Asp Pro Pro Pro Ala Ala Val Gln Phe Glu Gln Leu Ala Ala
65 70 75 80
Thr Asn His His Ala Met Ser Met Leu Arg Asp Tyr Tyr Gly Gln Gln
85 90 95
Tyr Pro Ala Glu Thr Tyr Leu Arg Gly Gly Pro Arg Thr Thr Thr Gly
100 105 110
Ser Ser Ser Leu Val Phe Gly Val Ala His Asp Asp Glu Ser Ala Ala
115 120 125
Tyr Asn Met Val Gly Pro Phe Val Glu Ser Ser Pro Thr Thr Arg Ala
130 135 140
Ala Gly Gly Gly Arg Lys Arg Asn Arg Gly Ser Arg Ala Ala Gly Gly
145 150 155 160
Pro Ala His Gly Gly Val Glu Lys Lys Glu Lys Gln Arg Arg Leu Arg
165 170 175
Leu Thr Glu Lys Tyr Thr Ala Leu Met Leu Leu Ile Pro Asn Arg Thr
180 185 190
Lys Glu Asp Arg Ala Thr Val Ile Ser Asp Ala Ile Glu Tyr Ile Gln
195 200 205
Glu Leu Gly Arg Thr Val Glu Glu Leu Thr Leu Leu Val Gly Lys Lys
210 215 220
Arg Arg Arg Asn Gly Ala Gly Glu His His Leu His Gln Gly Asp Val
225 230 235 240
Val Asp Ala Ala Pro Ala Val Gly Ala Ala Gly Glu Leu Val Leu Ala
245 250 255
Ala Glu Ser Ser Glu Gly Glu Val Gln Ala Pro Leu Ala Ala Leu Gln
260 265 270
Pro Ile Arg Ser Thr Tyr Ile Gln Arg Lys Ser Lys Glu Thr Phe Val
275 280 285
Asp Val Arg Ile Val Glu Asp Glu Val Asn Ile Lys Leu Thr Lys Arg
290 295 300
Arg Arg Asp Gly Cys Leu Ala Ala Ala Ser Arg Ala Leu Asp Asp Leu
305 310 315 320
Arg Leu Asp Leu Val His Leu Ser Gly Gly Lys Ile Gly Asp Cys His
325 330 335
Ile Tyr Met Phe Asn Thr Lys Ile His Gln Gly Ser Pro Val Phe Ala
340 345 350
Ser Ala Val Ala Ser Lys Leu Ile Glu Val Val Asp Glu Tyr
355 360 365
<210> 227
<211> 379
<212> БЕЛОК
<213> Sorghum bicolor
<400> 227
Met Tyr His Pro Gln Cys Glu Leu Leu Met Ala His Glu Ala Gln Asp
1 5 10 15
Leu Asp Ala Ala Gly Gln Pro His His Leu Ala Val Ser Gly Val Ala
20 25 30
Gly Ser Ile Pro Ala Glu Leu Ser Phe His Leu Leu His Ser Leu Asp
35 40 45
Ala Thr Ala Ala Val Asn Asn Ser Val Thr Pro Gln Ser Thr Ile Asp
50 55 60
Tyr Phe Leu Gly Val Gly Gly Ala Asp Pro His Gln Pro Ala Ala Leu
65 70 75 80
Gln Tyr Glu Pro Leu Pro Pro Pro Gly Gly His His Gln His Thr Met
85 90 95
Asn Met Leu Arg Asp Tyr Cys Ser Asn Gly Gly Gly Gly Gly His Tyr
100 105 110
Pro Thr Ala Glu Pro Tyr Leu Arg Gly Thr Arg Thr Gly Ala Leu Val
115 120 125
Phe Gly Ala Thr Asp Asp Asp Glu Ser Ala Ala Ala Tyr Met Pro Gly
130 135 140
Gly Pro Phe Val Glu Thr Ser Pro Pro Pro Arg Ala Thr Gly Gly Arg
145 150 155 160
Lys Arg Gly Arg Ala Leu Gly Gly Gly Phe His Ala Gly Leu Ala Asn
165 170 175
Gly Val Glu Lys Lys Glu Lys Gln Arg Arg Gln Arg Leu Thr Glu Lys
180 185 190
Tyr Thr Ala Leu Met His Leu Ile Pro Asn Val Thr Lys Pro Asp Arg
195 200 205
Ala Thr Val Ile Ser Asp Ala Ile Glu Tyr Ile Gln Glu Leu Gly Arg
210 215 220
Thr Val Glu Glu Leu Thr Leu Leu Val Glu Lys Lys Arg Arg Arg Arg
225 230 235 240
Glu Leu Gln Gly Asp Val Val Asp Ala Ala Pro Thr Ala Val Val Val
245 250 255
Ala Ala Ala Ala Thr Gly Gly Glu Ala Glu Ser Ser Glu Gly Glu Val
260 265 270
Ala Pro Pro Pro Pro Pro Pro Ala Ala Val Gln Arg Gln Pro Ile Arg
275 280 285
Ser Thr Tyr Ile Gln Arg Arg Ser Lys Asp Thr Ser Val Asp Val Arg
290 295 300
Ile Val Glu Glu Asp Val Asn Ile Lys Leu Thr Lys Arg Arg Arg Asp
305 310 315 320
Gly Cys Leu Ala Ala Ala Ser Arg Ala Leu Asp Asp Leu Arg Leu Asp
325 330 335
Leu Val His Leu Ser Gly Gly Lys Ile Gly Asp Cys His Ile Tyr Met
340 345 350
Phe Asn Thr Lys Ile His Lys Gly Ser Ser Val Phe Ala Ser Ala Val
355 360 365
Ala Ser Arg Leu Met Glu Val Val Asp Glu Tyr
370 375
<210> 228
<211> 379
<212> БЕЛОК
<213> Oryza sativa
<400> 228
Met Tyr His Pro Gln Cys Glu Leu Leu Met Pro Leu Glu Ser Leu Glu
1 5 10 15
Met Asp Val Gly Gln Ser His Leu Ala Ala Ala Val Ala Ala Ala Met
20 25 30
Pro Gly Glu Leu Asn Phe His Leu Leu His Ser Leu Asp Ala Ala Ala
35 40 45
Ala Ala Ala Ser Ser Thr Ala Ala Ser Ala Ser Ser Gln Pro Thr Val
50 55 60
Asp Tyr Phe Phe Gly Gly Ala Asp Gln Gln Pro Pro Pro Pro Ala Ala
65 70 75 80
Met Gln Tyr Asp Gln Leu Ala Ala Pro His His His Gln Thr Val Ala
85 90 95
Met Leu Arg Asp Tyr Tyr Gly Gly His Tyr Pro Pro Ala Ala Ala Ala
100 105 110
Ala Ala Ala Thr Glu Ala Tyr Phe Arg Gly Gly Pro Arg Thr Ala Gly
115 120 125
Ser Ser Ser Leu Val Phe Gly Pro Ala Asp Asp Glu Ser Ala Phe Met
130 135 140
Val Gly Pro Phe Glu Ser Ser Pro Thr Pro Arg Ser Gly Gly Gly Arg
145 150 155 160
Lys Arg Ser Arg Ala Thr Ala Gly Phe His Gly Gly Gly Pro Ala Asn
165 170 175
Gly Val Glu Lys Lys Glu Lys Gln Arg Arg Leu Arg Leu Thr Glu Lys
180 185 190
Tyr Asn Ala Leu Met Leu Leu Ile Pro Asn Arg Thr Lys Glu Asp Arg
195 200 205
Ala Thr Val Ile Ser Asp Ala Ile Glu Tyr Ile Gln Glu Leu Gly Arg
210 215 220
Thr Val Glu Glu Leu Thr Leu Leu Val Glu Lys Lys Arg Arg Arg Arg
225 230 235 240
Glu Met Gln Gly Asp Val Val Asp Ala Ala Thr Ser Ser Val Val Ala
245 250 255
Gly Met Asp Gln Ala Ala Glu Ser Ser Glu Gly Glu Val Met Ala Ala
260 265 270
Ala Ala Met Gly Ala Val Ala Pro Pro Pro Arg Gln Ala Pro Ile Arg
275 280 285
Ser Thr Tyr Ile Gln Arg Arg Ser Lys Glu Thr Phe Val Asp Val Arg
290 295 300
Ile Val Glu Asp Asp Val Asn Ile Lys Leu Thr Lys Arg Arg Arg Asp
305 310 315 320
Gly Cys Leu Ala Ala Ala Ser Arg Ala Leu Asp Asp Leu Arg Leu Asp
325 330 335
Leu Val His Leu Ser Gly Gly Lys Ile Gly Asp Cys His Ile Tyr Met
340 345 350
Phe Asn Thr Lys Ile His Ser Gly Ser Pro Val Phe Ala Ser Ala Val
355 360 365
Ala Ser Arg Leu Ile Glu Val Val Asp Glu Tyr
370 375
<210> 229
<211> 7
<212> БЕЛОК
<213> Искусственная
<220>
<223> Сигнал ядерной локализации из Rfr-bHLH кукурузы
<400> 229
Gly Arg Lys Arg Gly Arg Ala
1 5
<210> 230
<211> 6
<212> БЕЛОК
<213> Искусственная
<220>
<223> Сигнал ядерной локализации из Rf4-bHLH кукурузы
<400> 230
Lys Lys Arg Arg Arg Arg
1 5
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ГЕН-ВОССТАНОВИТЕЛЬ RF4 ДЛЯ ЦИТОПЛАЗМАТИЧЕСКОЙ МУЖСКОЙ СТЕРИЛЬНОСТИ (CMS) C-ТИПА КУКУРУЗЫ, МОЛЕКУЛЯРНЫЕ МАРКЕРЫ И ИХ ПРИМЕНЕНИЕ | 2011 |
|
RU2729107C2 |
| Участки генов и гены, ассоциированные с повышенной урожайностью у растений | 2016 |
|
RU2758718C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ УВЕЛИЧЕНИЯ УРОЖАЙНОСТИ НИЗКОРОСЛЫХ РАСТЕНИЙ ПУТЕМ МАНИПУЛЯЦИИ МЕТАБОЛИЗМА ГИББЕРЕЛЛИНА | 2017 |
|
RU2788379C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ УВЕЛИЧЕНИЯ УРОЖАЙНОСТИ НИЗКОРОСЛЫХ РАСТЕНИЙ ПУТЕМ МАНИПУЛЯЦИИ МЕТАБОЛИЗМА ГИББЕРЕЛЛИНА | 2017 |
|
RU2837282C2 |
| РАСТЕНИЯ С МОДИФИЦИРОВАННЫМИ ПРИЗНАКАМИ | 2017 |
|
RU2809117C2 |
| СВЯЗАННЫЙ С ФЕРТИЛЬНОСТЬЮ ГЕН ТаМS7 ПШЕНИЦЫ И СПОСОБ ЕГО ПРИМЕНЕНИЯ | 2017 |
|
RU2763468C2 |
| ГЕН-ВОССТАНОВИТЕЛЬ RF4 ДЛЯ ЦИТОПЛАЗМАТИЧЕСКОЙ МУЖСКОЙ СТЕРИЛЬНОСТИ (CMS) C-ТИПА КУКУРУЗЫ, МОЛЕКУЛЯРНЫЕ МАРКЕРЫ И ИХ ПРИМЕНЕНИЕ | 2011 |
|
RU2603005C2 |
| КОМПОЗИЦИИ ДЛЯ ИНДУКЦИИ ГАПЛОИДИИ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2816649C1 |
| СПОСОБЫ ПОЛУЧЕНИЯ ПРОМЫШЛЕННЫХ ПРОДУКТОВ ИЗ РАСТИТЕЛЬНЫХ ЛИПИДОВ | 2015 |
|
RU2743384C2 |
| ТРАНСГЕННОЕ РАСТЕНИЕ И СПОСОБ ЕГО ПОЛУЧЕНИЯ | 2018 |
|
RU2758722C2 |




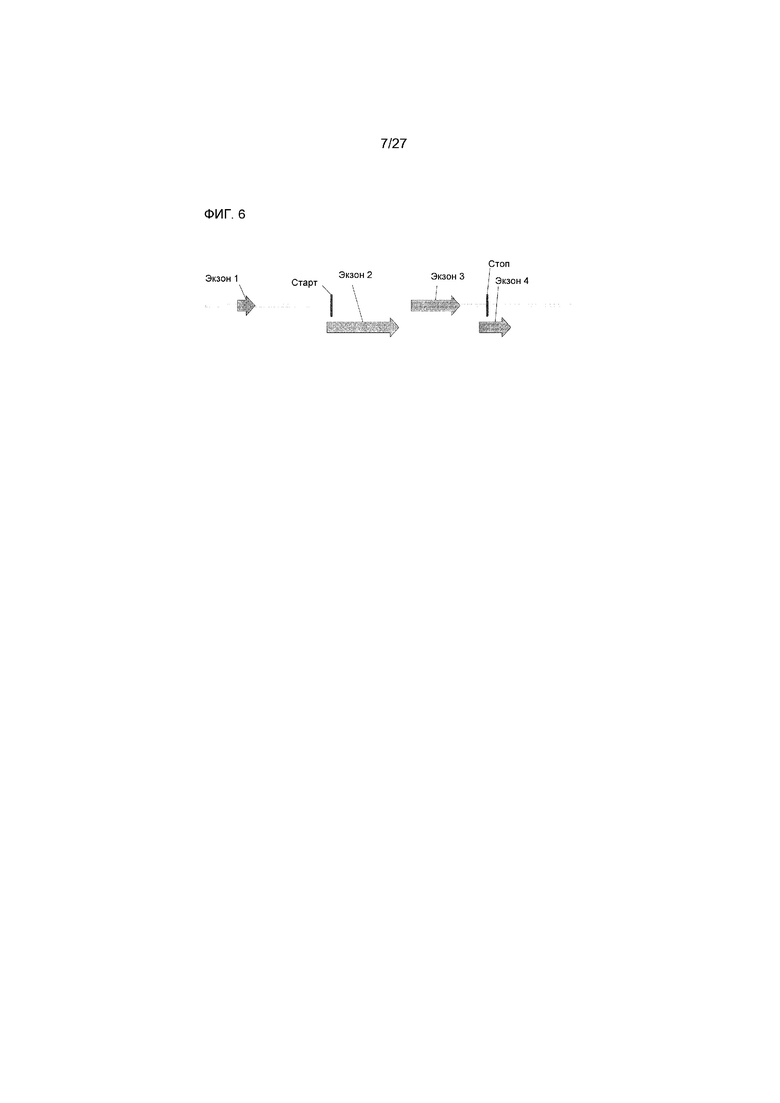

















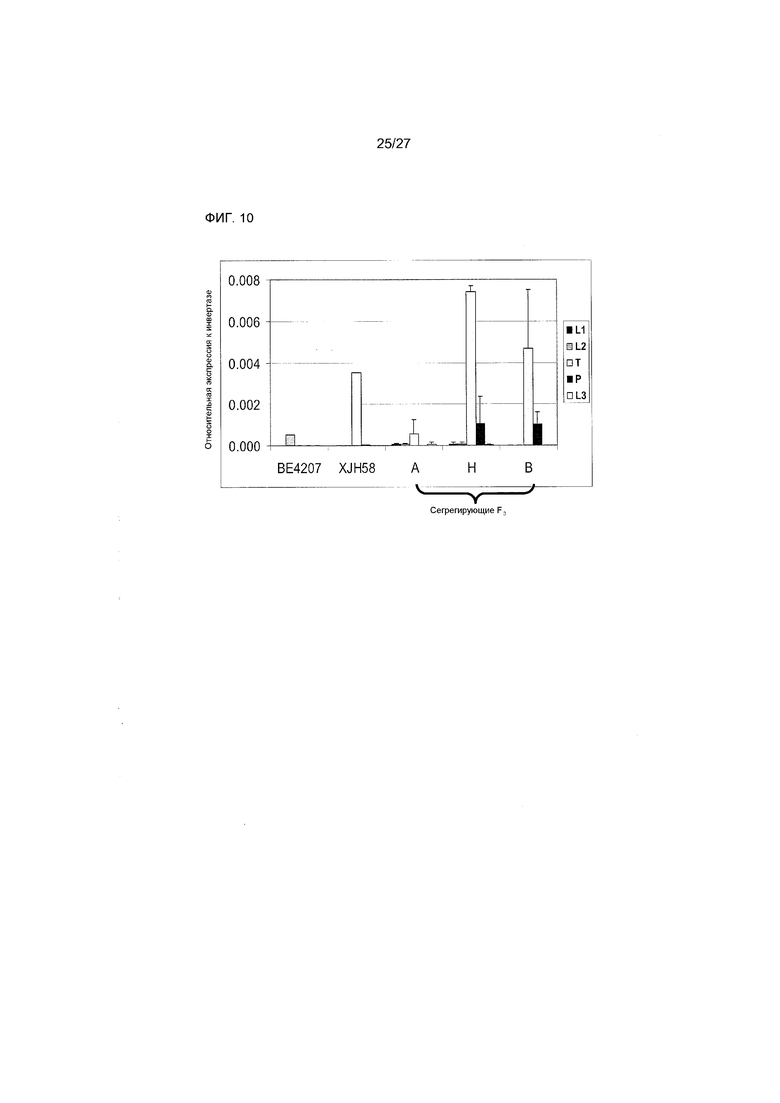


Изобретение относится к области биотехнологии, в частности к способу введения функционального гена-восстановителя Rf4 в растение кукурузы. Изобретение может быть эффективно использовано для восстановления цитоплазматической мужской стерильности в кукурузе. 6 з.п. ф-лы, 4 табл., 6 пр., 11 ил.
1. Способ введения функционального гена-восстановителя Rf4 в растение кукурузы, причем способ включает:
а) скрещивание родительского растения кукурузы, у которого отсутствует функциональный ген-восстановитель Rf4, с растением кукурузы, являющимся восстановителем мужской стерильности, имеющим признак способности отвечать на цитоплазматическую мужскую стерильность C-типа, обусловленный функциональным геном-восстановителем Rf4, с получением растений кукурузы F1;
b) выделение молекул нуклеиновой кислоты из одного или более растений-потомков кукурузы;
c) скрининг выделенных молекул нуклеиновых кислот на маркер, содержащий нуклеотидную последовательность, выбранную из группы, состоящей из SEQ ID NO:1-197, и маркеры, для которых указан полиморфизм ID № 1-106 в Таблице 3, где присутствие этого маркера указывает на присутствие функционального гена-восстановителя Rf4, отвечающего за цитоплазматическую мужскую стерильность C-типа растения кукурузы, и
d) отбор растения-потомка кукурузы, имеющего маркер, который указывает на присутствие функционального гена-восстановителя Rf4, таким образом осуществляя отбор растения-потомка, имеющего функциональный ген-восстановитель Rf4.
2. Способ по п.1, дополнительно включающий:
e) обратное скрещивание растения-потомка кукурузы, имеющего функциональный ген-восстановитель Rf4 с родительским растением кукурузы, у которого отсутствует функциональный ген-восстановитель Rf4 для получения растений-потомков кукурузы, созданных путем обратного скрещивания;
f) выделение молекул нуклеиновых кислот из одного или более растений-потомков кукурузы, созданных путем обратного скрещивания;
g) скрининг выделенных молекул нуклеиновых кислот на присутствие маркера, который указывает на присутствие функционального гена-восстановителя Rf4, отвечающего за цитоплазматическую мужскую стерильность C-типа растения кукурузы, с помощью секвенирования нуклеиновой кислоты или маркер-вспомогательной селекции;
h) отбор растения-потомка кукурузы, созданного путем обратного скрещивания и имеющего маркер, который указывает на присутствие функционального гена-восстановителя Rf4, таким образом осуществляя отбор растения-потомка кукурузы, созданного путем обратного скрещивания и имеющего функциональный ген-восстановитель Rf4; и
i) необязательно, повторение стадий обратного скрещивания e)-h) до момента, когда линия растений кукурузы, созданная путем обратного сращивания, будет содержать желаемые признаки родительского растения кукурузы и функциональный ген-восстановитель Rf4.
3. Способ по п. 1, где скрининг включает скрининг выделенных молекул нуклеиновой кислоты способом, включающим полимеразную цепную реакцию (ПЦР), на маркер, который указывает на присутствие функционального гена-восстановителя, отвечающего за цитоплазматическую мужскую стерильность C-типа растения кукурузы.
4. Способ по п. 2, где скрининг включает скрининг выделенных молекул нуклеиновой кислоты способом, включающим полимеразную цепную реакцию (ПЦР), на маркер, который указывает на присутствие функционального гена-восстановителя, отвечающего за цитоплазматическую мужскую стерильность C-типа растения кукурузы.
5. Способ по п. 1, где скрининг включает секвенирование ДНК на присутствие Rf4 последовательности SEQ ID NO:220.
6. Способ по п. 2, где скрининг включает секвенирование ДНК на присутствие Rf4 последовательности SEQ ID NO:220.
7. Способ по любому из пп. 1-6, где отобранное потомство или линия растений-потомков кукурузы, созданная путем обратного скрещивания, имеющие функциональный ген-восстановитель Rf4, используются для восстановления цитоплазматической мужской стерильности в кукурузе, причем способ включает:
скрещивание женского стерильного растения кукурузы, имеющего признак цитоплазматической мужской стерильности C-типа, с растением кукурузы из отобранного потомства или линии растений-потомков кукурузы, созданных путем обратного скрещивания, являющимся восстановителем мужской стерильности, содержащим средства для восстановления фертильности кукурузы CMS-C, с получением растений кукурузы F1;
идентификацию растения кукурузы F1, содержащего средства для восстановления фертильности кукурузы CMS-C; и
размножение идентифицированного растения кукурузы F1, тем самым восстанавливая мужскую стерильность в кукурузе.
| KOHLS S | |||
| et al., Fine-Mapping of Rf4, a Major Restorer-of-Fertility Gene for c-Type Cytoplasmic Male Sterility in Maize, in Program and Abstracts of the 52nd Annual Maize Genetics Conference, March 2010 | |||
| PAUL H | |||
| et al., Duplications Complicate Genetic Mapping of Rf4, a Restorer Gene for cms-C Cytoplasmic Male Sterility in Corn, Published in Crop |
Авторы
Даты
2021-05-18—Публикация
2011-09-26—Подача