ОБЛАСТЬ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к фармацевтической композиции для предупреждения или лечения заболеваний кожи, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом, имеющим происхождение из выделенного виментина. Конкретно, настоящее изобретение относится к фармацевтической композиции, косметической композиции, квазилекарственной композиции и препарату для накожного применения для предупреждения или лечения заболеваний кожи, содержащим, в качестве активного ингредиента, антитело, специфично связывающееся с пептидом SEQ ID NO: 1, или фрагмент, связывающийся с указанным пептидом, и к способу предупреждения или лечения заболеваний кожи, их терапевтическому применению и их применению для изготовления лекарственного средства, косметических средств, квазилекарственных средств, препаратов для накожного применения и так далее.
ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ
Заболевание кожи относится ко всем отклонениям от нормы, встречающимся в коже животных, включая людей. По мере роста индустриализации в современном обществе возникают различные ранее не встречавшиеся заболевания кожи, что обусловлено различными вредными веществами. В частности, со временем пролиферация или метаболизм клеток кожи ослабевают, результатом чего становится естественное старение, приводящее к снижению эластичности тканей кожи или появлению морщин. Кроме того, различные поражения кожи могут возникать при длительном воздействии прямого солнечного света, ожогах, ссадинах и так далее, что может приводить к повреждению здоровой кожи. Такие поражения кожи, вызванные естественным старением или травмами, приводят к снижению защитной функции кожи, которую она выполняет как часть человеческого тела, непосредственно контактирующая с внешней средой, и, посредством этого, к инфицированию вирусами, микроорганизмами, бактериями и так далее, способствуя таким образом развитию различных заболеваний кожи.
Атопический дерматит, типичное заболевание, конкретная этиология которого не установлена, представляет собой хроническое воспалительное заболевание кожи, возникающее чаще всего в младенческом или детском возрасте, выраженность симптомов которого повторно уменьшается и возрастает, при этом его обострения могут быть вызваны инфекциями, эмоциональным стрессом, сезонными и климатическими изменениями, раздражением и аллергенами. Несмотря на признание атопического дерматита наследственным заболеванием, связанным с иммунологическими отклонениями, его конкретная этиология пока не установлена. Причины других заболеваний кожи, таких как псориаз, простой хронический лишай и так далее, также неизвестны.
В лечении заболеваний кожи, таких как атопический дерматит, псориаз, простой хронический лишай, розовый лишай и так далее, конкретная этиология которых пока не установлена, чаще всего используют противовоспалительные агенты и противовирусные агенты, состоящие из химических лекарственных средств или соединений, имеющих происхождение из растительных экстрактов или натуральных продуктов. Например, в патенте Кореи №10-1741281 раскрыта «фармацевтическая композиция, содержащая агонист А3-аденозиновых рецепторов (IB-MECA/CF-101) для лечения псориаза», и в публикации заявки на патент Кореи №10-2017-0041149 раскрыта «композиция для улучшения состояния атопичной кожи, содержащая ферментативно гидролизованный экстракт Aronia и экстракт Orostachys japonicus.
Тем не менее, вероятность полного излечения таких заболеваний кожи этими способами лечения довольно мала, и, поскольку эти способы не являются терапевтическим агентом, действующим только на ткань кожи и клетки кожи пораженной области, эффективность такого лечения весьма низкая, а вероятность побочных эффектов относительно высока.
ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Техническая проблема
В этих обстоятельствах авторы настоящего изобретения приложили значительные усилия для решения проблем, описанных выше. В результате, была подтверждена возможность эффективного лечения различных заболеваний кожи, конкретная этиология которых пока не ясна, при нанесении вещества, специфично связывающегося с пептидом, имеющим происхождение из виментина, конкретно, антитела, специфично связывающегося с пептидом SEQ ID NO: 1, на ткань кожи пациентов, страдающих от этих заболеваний кожи, посредством чего настоящее изобретение было завершено.
Техническое решение
Одной задачей настоящего изобретения является предложение фармацевтической композиции для предупреждения или лечения заболеваний кожи, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1.
Другой задачей настоящего изобретения является предложение косметической композиции для предупреждения или облегчения заболеваний кожи, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1.
Еще одной задачей настоящего изобретения является предложение квазилекарственной композиции для предупреждения или облегчения заболеваний кожи, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1.
Еще одной задачей настоящего изобретения является предложение препарата для накожного применения для предупреждения или облегчения заболеваний кожи, содержащего, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1.
Еще одной задачей настоящего изобретения является предложение способа предупреждения или лечения заболеваний кожи, включающего введение композиции, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1, субъекту.
Еще одной задачей настоящего изобретения является предложение применения композиции для предупреждения или лечения заболеваний кожи, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1.
Еще одной задачей настоящего изобретения является предложение применения композиции, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1, для изготовления лекарственного средства для предупреждения или лечения заболеваний кожи.
Еще одной задачей настоящего изобретения является предложение применения композиции, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1, для изготовления косметических средств, квазилекарственных средств или препаратов для накожного применения для облегчения заболеваний кожи.
Полезные эффекты
Вещество, специфично связывающееся с пептидом SEQ ID NO: 1, конкретнее, антитело, специфично связывающееся с пептидом SEQ ID NO: 1, или фрагмент, связывающийся с указанным пептидом, позволяют эффективно предупреждать и лечить заболевания кожи, сопровождающиеся экспрессией пептида, имеющего происхождение из виментина, и, таким образом, могут быть эффективно использованы в качестве терапевтических агентов применительно к различным заболеваниям кожи, конкретная этиология которых пока не ясна.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
На ФИГ. 1 представлена схематическая диаграмма вектора для получения химерного VSF.
На ФИГ. 2 представлена схематическая диаграмма химерного VSF.
На ФИГ. 3 представлена диаграмма, подтверждающая экспрессию химерного VSF.
На ФИГ. 4 представлена диаграмма, на которой показана последовательность ДНК scFv VSF.
На ФИГ. 5 представлена схематическая диаграмма, на которой показано клонирование scFv VSF в вектор. На ФИГ. 5 linker обозначает линкер; pelB coding sequence обозначает pelB кодирующая последовательность; His-Tag обозначает His-метка.
На ФИГ. 6 представлена диаграмма, подтверждающая scFV VSF после очистки.
На ФИГ. 7 представлена схематическая диаграмма вектора для получения гуманизированного антитела hzVSF.
На ФИГ. 8 представлена схематическая диаграмма гуманизированного антитела hzVSF по настоящему изобретению.
На ФИГ. 9 представлена диаграмма, на которой показана экспрессия гуманизированного антитела hzVSF.
На ФИГ. 10 представлена диаграмма, на которой показаны результаты электрофореза в полиакриламидном геле с додецилсульфатом натрия (SDS-PAGE) в восстанавливающих и невосстанавливающих условиях, подтвердившие физические свойства гуманизированного антитела hzVSF_var13.
На ФИГ. 11 представлена диаграмма, на которой показаны результаты жидкостной хроматографии/масс-спектрометрии (LC/MS), подтвердившие физические свойства гуманизированного антитела hzVSF_var13.
На ФИГ. 12 представлена диаграмма, на которой показаны результаты эксклюзионной высокоэффективной жидкостной хроматографии (SEC-HPLC), подтвердившие физические свойства гуманизированного антитела hzVSF_var13.
На ФИГ. 13 представлена диаграмма, на которой показано изоэлектрическое фокусирование (IF), подтвердившее физические свойства гуманизированного антитела hzVSF_var13.
На ФИГ. 14 представлена диаграмма, на которой показано количество доноров с пролиферацией Т-клеток в ответ на KLH, hzVSF_var12 и hzVSF_var13 из 51 донора крови.
На ФИГ. 15 представлена диаграмма, на которой показана степень пролиферации Т-клеток у 51 донора в ответ на hzVSF_var12 и hzVSF_var13, являющиеся типичными вариантами гуманизированного антитела hzVSF.
На ФИГ. 16 представлена диаграмма, на которой показана пролиферация Т-клеток, индуцированная KLH, hzVSF_var12 и hzVSF_var13, в виде средних значений индекса стимуляции (SI).
На ФИГ. 17 представлена диаграмма, на которой показаны результаты по взаимодействию нормальных человеческих тканей и VSF. На ФИГ. 17 Skin обозначает кожа; Breast обозначает молочная железа; Spleen обозначает селезенка; Rectum обозначает прямая кишка; Kidney, cortex обозначает почка, корковое вещество; Kidney, medulla обозначает почка, мозговое вещество; Gallbladder обозначает желчный пузырь; Pancreas обозначает поджелудочная железа; Tonsil обозначает миндалина; Prostate обозначает предстательная железа; Testis обозначает яичко; Uterine cervix обозначает шейка матки; Stomach, antrum обозначает желудок, антральный отдел; Stomach, fundus обозначает желудок, фундальный отдел; Small bowel обозначает тонкая кишка; Myometrium обозначает миометрий; Placenta обозначает плацента; Adrenal gland обозначает надпочечник; Skeletal muscle обозначает скелетная мышца; Lung обозначает легкое; Salivary gland обозначает слюнная железа; Cerebrum обозначает головной мозг; Cerebellum обозначает мозжечок; Lymph node обозначает лимфатический узел.
На ФИГ. 18 представлена диаграмма, подтверждающая экспрессию VR при различных заболеваниях кожи.
На ФИГ. 19 представлена диаграмма, подтверждающая экспрессию VR при розовом лишае, опоясывающем герпесе, простом хроническом лишае, простом герпесе, нумуллярной экземе и контагиозном моллюске.
На ФИГ. 20 представлена диаграмма, подтверждающая экспрессию VR при псориазе, простых бородавках, атопическом дерматите и аллергическом контактном дерматите.
На ФИГ. 21 представлена диаграмма, подтверждающая терапевтическую эффективность VSF у пациента 1 с псориазом.
На ФИГ. 22 представлена диаграмма, подтверждающая терапевтическую эффективность VSF у пациента 2 с псориазом.
На ФИГ. 23 представлена диаграмма, подтверждающая терапевтическую эффективность VSF у пациента 1 с простым хроническим лишаем.
На ФИГ. 24 представлена диаграмма, подтверждающая терапевтическую эффективность VSF у пациента 2 с простым хроническим лишаем.
На ФИГ. 25 представлена диаграмма, подтверждающая терапевтическую эффективность VSF у пациента 3 с простым хроническим лишаем.
На ФИГ. 26 представлена диаграмма, подтверждающая терапевтическую эффективность VSF у пациента с нумуллярной экземой.
На ФИГ. 27 представлена диаграмма, на которой показаны значения индекса площади и тяжести экземы (eczema area and severity index, EASI), позволяющего оценить эритему, отек/бугорки/папулы, осаднение и лихенификацию у пациентов с простым хроническим лишаем, нумуллярной экземой, экземой, атопическим дерматитом и дерматитом, после нанесения VSF, и значения индекса площади и тяжести псориаза (psoriasis area severity index, PASI), позволяющего оценить эритему, утолщение и шелушение кожи у пациентов с псориазом.
На ФИГ. 28 представлена диаграмма, на которой показана визуальная аналоговая шкала (visual analogue scale, VAS) у пациентов с простым хроническим лишаем, нумуллярной экземой, экземой, атопическим дерматитом, дерматитом и псориазом после нанесения VSF.
ПОДРОБНОЕ ОПИСАНИЕ ПРЕДПОЧТИТЕЛЬНЫХ ВОПЛОЩЕНИЙ
Конкретные подробности настоящего изобретения могут быть объяснены следующим образом. В то же время, объяснения и воплощения, раскрытые в настоящем изобретении, могут быть применены к другим объяснениям и воплощениям, соответственно. То есть, объем настоящего изобретения включает все комбинации различных элементов, раскрытых здесь. Кроме того, объем настоящего изобретения не следует ограничивать конкретными описаниями, приведенными ниже.
Для решения указанных выше задач согласно одному аспекту настоящего изобретения предложена фармацевтическая композиция для предупреждения или лечения заболеваний кожи, содержащая, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом, имеющим происхождение из виментина.
Более конкретно, согласно настоящему изобретению предложена фармацевтическая композиция для предупреждения или лечения заболеваний кожи, содержащая, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1.
При использовании здесь виментин, представляющий собой белок, кодируемый геном VIM, выполняет функцию поддержания и фиксации положения внутриклеточных органелл, и известно, что он вовлечен главным образом в поддержание цитоскелета, транспорт белков и передачу клеточных сигналов. В настоящем изобретении было подтверждено, что, несмотря на отсутствие внеклеточной экспрессии виментина в нормальных клетках, он экспрессирован внеклеточно в тканях кожи пациентов с различными заболеваниями кожи, конкретная этиология которых пока не ясна, и, кроме того, что нанесение вещества, способного связываться с пептидом, имеющим происхождение из виментина, такого как антитело, специфичное в отношении этого пептида, на ткани кожи пациентов позволяет эффективно лечить различные заболевания кожи.
Конкретно, пептид, имеющий происхождение из виментина, может представлять собой пептид SEQ ID NO: 1.
В настоящем изобретении «вещество, специфично связывающееся с пептидом, имеющим происхождение из виментина», представляющее собой вещество, имеющее аффинность связывания в отношении пептида, имеющего происхождение из виментина, может, конкретно, представлять собой вещество, специфично связывающееся с пептидом SEQ ID NO: 1.
В настоящем изобретении выделенный пептид SEQ ID NO: 1 соответствует аминокислотной последовательности виментина в аминокислотных положениях 142-294, и указанный пептид может содержать не только указанную выше аминокислотную последовательность, но также любую аминокислотную последовательность, на 80% или более, предпочтительно на 90% или более, более предпочтительно на 95% или более и еще более предпочтительно на 97% или более гомологичную указанной выше последовательности, при условии, что с ним могут связываться антитело по настоящему изобретению или его фрагмент.
Конкретнее, вещество, специфично связывающееся с пептидом SEQ ID NO: 1, может представлять собой антитело, специфично связывающееся с пептидом SEQ ID NO: 1, или фрагмент, связывающийся с указанным пептидом, соединение, пептид или аптамер пептида. Конкретнее, вещество может представлять собой антитело, специфично связывающееся с пептидом SEQ ID NO: 1, или фрагмент, связывающийся с указанным пептидом, но не ограничено ими.
Конкретно, согласно настоящему изобретению предложена фармацевтическая композиция для предупреждения или лечения заболеваний кожи, содержащая, в качестве активного ингредиента, антитело, специфично связывающееся с пептидом SEQ ID NO: 1, или фрагмент, связывающийся с указанным пептидом.
Примеры антитела могут включать мышиные антитела, химерные антитела или гуманизированные антитела, но не ограничены ими.
Гуманизированное антитело или фрагмент, связывающиеся с пептидом по настоящему изобретению, обладают преимуществом ингибирования реакции организма человека против мышиных антител (human anti-mouse antibody reaction, HAMA) и, в то же время, сохраняют исходную аффинность и специфичность мышиного антитела, благодаря переносу гипервариабельного участка вариабельной области мышиного моноклона или моноклонального антитела, непосредственно связывающегося с антигеном, на каркас человеческого антитела. Кроме того, иммуногенность гуманизированных антител по настоящему изобретению снижена посредством деиммунизации, и поэтому они могут быть использованы в качестве агента, безопасного при введении людям, благодаря существенному снижению их иммуногенности. То есть, гуманизированные антитела по настоящему изобретению могут действовать на клетки-мишени более эффективно, благоприятным образом взаимодействуя с иммунной системой человека и, в то же время, отвечая и влияя на клетки, на поверхности мембраны которых представлена пептидная область SEQ ID NO: 1, например, предупреждая комплемент-зависимую цитотоксичность (complement-dependent cytotoxicity, CDC) или антителозависимую клеточно-опосредованную цитотоксичность (antibody-dependent cell-mediated cytotoxicity, ADCC). Кроме этого, гуманизированные антитела по настоящему изобретению обладают тем преимуществом, что иммунная система человека не распознает эти гуманизированные антитела как белки чужеродного происхождения, благодаря их сниженной иммуногенности. Кроме этого, гуманизированные антитела по настоящему изобретению также обладают тем преимуществом, что периоды полувыведения этих гуманизированных антител из кровеносной системы человека близки к периодам полувыведения естественных антител, даже при введении лекарственного средства в меньшей дозе и с меньшей частотой.
В настоящем изобретении мышиные антитела, специфично связывающиеся с выделенным пептидом SEQ ID NO: 1, могут, вместе взятые, быть названы «мышиным вирусоподавляющим фактором (mouse virus suppressing factor, mVSF)»; химерные антитела, «химерным вирусоподавляющим фактором (chimeric virus suppressing factor, chVSF)»; и гуманизированные антитела, «гуманизированным вирусоподавляющим фактором (humanized virus suppressing factor, hzVSF)». При использовании здесь термин «гуманизированное антитело hzVSF» или «его варианты» могут быть использованы взаимозаменяемо, и hzVSF может быть использован взаимозаменяемо с hzVSF дикого типа (hzVSF_wt) и вариантом hzVSF (например, обозначенными как hzVSF_var1, hzVSF_v1, hzVSF_1 и так далее).
Выделенный пептид SEQ ID NO: 1 представляет собой антигенную область, содержащую эпитоп, и он может представлять собой аминокислотную последовательность виментина в аминокислотных положениях 142-211 или в аминокислотных положениях 211-294 при условии, что пептид может демонстрировать функцию, сходную с функцией пептида по настоящему изобретению, связываясь с антителом или пептид-связывающим фрагментом. Кроме того, очевидно, что объем настоящего изобретения может включать любую аминокислотную последовательность, имеющую такую гомологию, описанную выше, несмотря на то, что эта последовательность может иметь частичную делецию, модификацию, замену или добавление. Виментин, представляющий собой белок, кодируемый геном VIM, выполняет функции поддержания и фиксации положения внутриклеточных органелл, и известно, что он вовлечен главным образом в поддержание цитоскелета, транспорт белков и передачу клеточных сигналов. Также известно, что виментин используют в качестве онкомаркера, тем не менее, неизвестно, можно ли использовать антитела, способные связываться с виментином, для лечения заболеваний кожи.
Антитело, специфично связывающееся с выделенным пептидом SEQ ID NO: 1 по настоящему изобретению, или фрагмент, связывающийся с указанным пептидом, специфично отвечают на клетки кожи пациентов с заболеваниями кожи, и антитело или фрагмент, связывающиеся с указанным пептидом, связываются с рецепторами вирусоподавляющего фактора (VSF), представленными на поверхности клеток тканей кожи или клеток кожи в области, пораженной этими заболеваниями. Поэтому антитело или фрагмент, связывающиеся с указанным пептидом по настоящему изобретению, могут быть эффективно использованы в области предупреждения или лечения различных заболеваний кожи, конкретная этиология которых пока не ясна.
Конкретно, антитело или фрагмент, связывающиеся с указанным пептидом, могут представлять собой антитело или фрагмент, специфично связывающиеся с аминокислотным остатком в положении 9, 45, 54, 76, 94 или 129 пептида SEQ ID NO: 1, и, конкретнее, антитело или фрагмент, специфично связывающиеся с аминокислотными остатками в положениях 9, 45, 54, 76, 94 и 129 пептида SEQ ID NO: 1, но не ограничены ими, при условии, что антитело или фрагмент могут специфично связываться с выделенным пептидом SEQ ID NO: 1.
При использовании здесь термин «антитело» иммунологически относится к молекуле белка, выполняющего роль лиганда, специфично распознающего антиген, включая молекулу иммуноглобулина, обладающую реактивностью в отношении конкретного антигена, и может включать все из поликлонального антитела, моноклонального антитела, целого антитела и фрагмента антитела. Кроме того, термин «антитело» может включать химерное антитело (например, гуманизированное мышиное антитело), бивалентную или биспецифичную молекулу (например, биспецифичное антитело), диатело, триатело и тетратело. Кроме того, термин «антитело» может включать одноцепочечное антитело, обладающее аффинностью связывания в отношении FcRn, scAb, производное константной области антитела, искусственное антитело на основе белкового каркаса. Структура целого антитела состоит из двух полноразмерных легких цепей и двух полноразмерных тяжелых цепей, где каждая легкая цепь связана с тяжелой цепью дисульфидной связью. Целое антитело включает IgA, IgD, IgE, IgM и IgG, и подтипы IgG включают IgG1, IgG2, IgG3 и IgG4. При использовании здесь термины «фрагмент», «фрагмент, связывающийся с пептидом», и «фрагмент антитела» относятся к любому фрагменту антител по настоящему изобретению, обладающему антигенсвязывающей активностью этих антител, и эти термины могут быть использованы взаимозаменяемо. Примеры фрагментов антител включают одноцепочечное антитело, Fd, Fab, Fab', F(ab')2, dsFv или scFv, но не ограничены ими. Fd относится к части тяжелой цепи, включенной в Fab-фрагмент. Структура Fab состоит из вариабельных областей тяжелой цепи и легкой цепи, константных областей легкой цепи и первой константной области тяжелой цепи (домена СН1) и имеет один антигенсвязывающий домен. Fab' отличается от Fab тем, что у Fab' есть шарнирная область, содержащая по меньшей мере один цистеиновый остаток, на С-конце домена СН1 тяжелой цепи. Антитело F(ab')2 возникает при образовании дисульфидной связи между цистеиновыми остатками шарнирной области Fab'. При использовании здесь термин «вариабельный фрагмент (Fv)» относится к минимальному фрагменту антитела, имеющему только вариабельную область тяжелой цепи и вариабельную область легкой цепи. Дисульфидно стабилизированный Fv (dsFv) характеризуется тем, что вариабельная область тяжелой цепи и вариабельная область легкой цепи связаны дисульфидной связью, и одноцепочечный Fv (scFv) характеризуется тем, что вариабельная область тяжелой цепи и вариабельная область легкой цепи обычно связаны ковалентной связью через линкер. Эти фрагменты антител могут быть получены с использованием протеазы (например, рестрикционное расщепление целого антитела папаином позволяет получить Fab, в то время как расщепление целого антитела пепсином позволяет получить F(ab')2-фрагменты) и могут, предпочтительно, быть получены с применением генетической рекомбинантной технологии.
Конкретно, фрагмент, связывающийся с пептидом, может представлять собой Fab, Fab', F(ab')2, scFv, dsFv, ds-scFv, их димеры, минитела, диатела, мультимеры или фрагменты биспецифичных антител.
При использовании здесь термин «моноклональное антитело» относится к молекуле антитела, состоящей из одной молекулы, полученной из по существу одной и той же группы антител, и моноклональное антитело демонстрирует одну специфичность и аффинность связывания в отношении определенного эпитопа.
Обычно иммуноглобулины имеют тяжелые цепи и легкие цепи, и каждая тяжелая цепь и легкая цепь содержит константную область и вариабельную область (также известные как домены). Вариабельные области легкой цепи и тяжелой цепи содержат три высоко вариабельные области, называемые гипервариабельными участками (complementarity-determining regions, CDR), и четыре каркасные области (framework regions, FR). Основной ролью CDR является связывание с эпитопом антигена. В каждой цепи CDR называют последовательно CDR1, CDR2 и CDR3, обычно начиная с N-конца, и их определяют по цепи, в которой расположен конкретный CDR.
Кроме того, когда антитело по настоящему изобретению содержит константную область, оно может содержать константную область, имеющую происхождение из IgG, IgA, IgD, IgE и IgM, или их комбинацию или гибрид.
При использовании здесь термин «комбинация» относится к образованию связи между полипептидом, кодирующим одноцепочечную константную область иммуноглобулина одного происхождения, и одноцепочечным полипептидом другого происхождения при образовании димера или мультимера. Например, димер или мультимер могут быть образованы двумя или более константными областями, выбранными из группы, состоящей из константных областей IgG, IgA, IgD, IgE и IgM.
При использовании здесь термин «гибрид» относится к присутствию последовательностей, соответствующих двум или более константным областям тяжелой цепи иммуноглобулина разного происхождения в одноцепочечной константной области тяжелой цепи иммуноглобулина, и, например, может быть возможен гибрид, состоящий из одного-четырех доменов, выбранных из группы, состоящей из CH1, СН2, СН3 и СН4 IgG, IgA, IgD, IgE и IgM.
Гуманизированное антитело по настоящему изобретению может быть гуманизировано на основе человеческого иммуноглобулина у4, однако гуманизация не ограничена этим вариантом, и оно может обладать тем преимуществом, что оно не приводит к комплемент-зависимой цитотоксичности (CDC), благодаря отсутствию связывания комплемента.
Гуманизированное антитело или фрагмент, связывающиеся с указанным пептидом, могут содержать: вариабельную область тяжелой цепи, содержащую CDR1 тяжелой цепи SEQ ID NO: 2, CDR2 тяжелой цепи SEQ ID NO: 3 или SEQ ID NO: 14 (где треонин, аминокислота 9 SEQ ID NO: 3, заменен аспарагиновой кислотой) и CDR3 тяжелой цепи SEQ ID NO: 4 или SEQ ID NO: 15 (где треонин, аминокислота 4 SEQ ID NO: 4, заменен аспарагином); и вариабельную область легкой цепи, содержащую CDR1 легкой цепи SEQ ID NO: 5, CDR2 легкой цепи SEQ ID NO: 6, SEQ ID NO: 16 (где треонин, аминокислота 3 SEQ ID NO: 6, заменен аспарагиновой кислотой), SEQ ID NO: 17 (где треонин, аминокислота 3 SEQ ID NO: 6, заменен аспарагиновой кислотой; и аланин, аминокислота 6 SEQ ID NO: 6, заменен глицином) или SEQ ID NO: 18 (где треонин, аминокислота 3 SEQ ID NO: 6, заменен аспарагиновой кислотой; лейцин, аминокислота 5 SEQ ID NO: 6, заменен аргинином; и аланин, аминокислота 6 SEQ ID NO: 6, заменен глицином) и CDR3 легкой цепи SEQ ID NO: 7 или SEQ ID NO: 19 (где серии, аминокислота 6 SEQ ID NO: 7, заменен треонином).
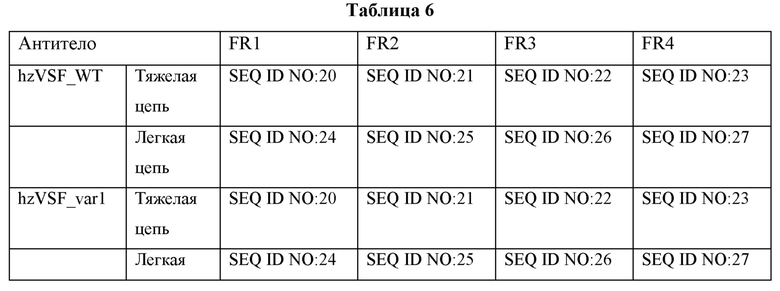
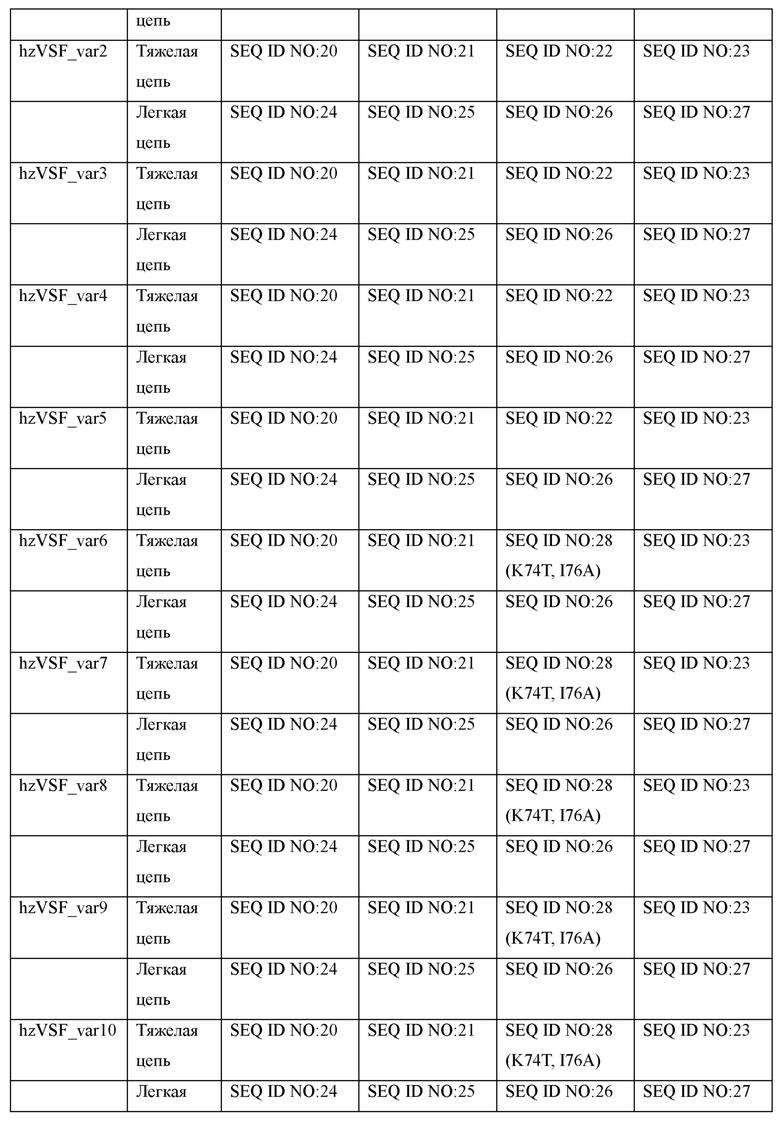
Кроме того, гуманизированное антитело или фрагмент, связывающиеся с указанным пептидом, могут содержать человеческую каркасную область (FR) и могут представлять собой человеческий иммуноглобулин-гамма SEQ ID NO: 29, SEQ ID NO: 30 и SEQ ID NO: 31 или вариабельную область тяжелой цепи, содержащую каркасную область 1 (FR1) тяжелой цепи SEQ ID NO: 20, FR2 тяжелой цепи SEQ ID NO: 21, FR3 тяжелой цепи SEQ ID NO: 22 или SEQ ID NO: 28 (где лизин, аминокислота 8 SEQ ID NO: 22, заменен треонином; и изолейцин, аминокислота 10 SEQ ID NO: 22, заменен аланином) и FR4 тяжелой цепи SEQ ID NO: 23, и вариабельную область легкой цепи, содержащую FR1 легкой цепи SEQ ID NO: 24, FR2 легкой цепи SEQ ID NO: 25, FR3 легкой цепи SEQ ID NO: 26 и FR4 легкой цепи SEQ ID NO: 27, но гуманизированное антитело или фрагмент не ограничены указанными вариантами.
Конкретно, гуманизированное антитело или фрагмент, связывающиеся с указанным пептидом, могут содержать:
(a) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 3 и SEQ ID NO: 4, соответственно; и CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 6 и SEQ ID NO: 7, соответственно; (b) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 3 и SEQ ID NO: 4, соответственно; и CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 16 и SEQ ID NO: 7, соответственно; (с) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 3 и SEQ ID NO: 4, соответственно; и CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 17 и SEQ ID NO: 7, соответственно; (d) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 3 и SEQ ID NO: 4, соответственно; и CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 18 и SEQ ID NO: 7, соответственно; (е) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 3 и SEQ ID NO: 4, соответственно; и CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 18 и SEQ ID NO: 19, соответственно; (f) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 14 и SEQ ID NO: 4, соответственно; и CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 6 и SEQ ID NO: 7, соответственно; (g) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 3 и SEQ ID NO: 4, соответственно; FR1, FR2, FR3 и FR4 тяжелой цепи SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 28 и SEQ ID NO: 23, соответственно; CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 6 и SEQ ID NO: 7, соответственно; и FR1, FR2, FR3 и FR4 легкой цепи SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26 и SEQ ID NO: 27, соответственно; (h) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 14 и SEQ ID NO: 4, соответственно; FR1, FR2, FR3 и FR4 тяжелой цепи SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 28 и SEQ ID NO: 23, соответственно; CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 6 и SEQ ID NO: 7, соответственно; и FR1, FR2, FR3 и FR4 легкой цепи SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26 и SEQ ID NO: 27, соответственно; (i) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 14 и SEQ ID NO: 15, соответственно; FR1, FR2, FR3 и FR4 тяжелой цепи SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 28 и SEQ ID NO: 23, соответственно; CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 6 и SEQ ID NO: 7, соответственно; и FR1, FR2, FR3 и FR4 легкой цепи SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26 и SEQ ID NO: 27, соответственно; (j) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 14 и SEQ ID NO: 4, соответственно; FR1, FR2, FR3 и FR4 тяжелой цепи SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 28 и SEQ ID NO: 23, соответственно; CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 18 и SEQ ID NO: 7, соответственно; и FR1, FR2, FR3 и FR4 легкой цепи SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26 и SEQ ID NO: 27, соответственно; (k) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 14 и SEQ ID NO: 15, соответственно; FR1, FR2, FR3 и FR4 тяжелой цепи SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 28 и SEQ ID NO: 23, соответственно; CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 18 и SEQ ID NO: 7, соответственно; и FR1, FR2, FR3 и FR4 легкой цепи SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26 и SEQ ID NO: 27, соответственно; (l) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 14 и SEQ ID NO: 4, соответственно; FR1, FR2, FR3 и FR4 тяжелой цепи SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 28 и SEQ ID NO: 23, соответственно; CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 18 и SEQ ID NO: 19, соответственно; и FR1, FR2, FR3 и FR4 легкой цепи SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26 и SEQ ID NO: 27, соответственно; (m) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 14 и SEQ ID NO: 15, соответственно; FR1, FR2, FR3 и FR4 тяжелой цепи SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 28 и SEQ ID NO: 23, соответственно; CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 18 и SEQ ID NO: 19, соответственно; и FR1, FR2, FR3 и FR4 легкой цепи SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26 и SEQ ID NO: 27, соответственно; и (n) CDR1 тяжелой цепи, CDR2 тяжелой цепи и CDR3 тяжелой цепи SEQ ID NO: 2, SEQ ID NO: 14 и SEQ ID NO: 4, соответственно; и CDR1 легкой цепи, CDR2 легкой цепи и CDR3 легкой цепи SEQ ID NO: 5, SEQ ID NO: 16 и SEQ ID NO: 7, соответственно.
Антитело (а) может содержать hzVSF_WT, антитело (b) может содержать hzVSF_var1, антитело (с) может содержать hzVSF_var2, антитело (d) может содержать hzVSF_var3, антитело (е) может содержать hzVSF_var4, антитело (f) может содержать hzVSF_var5, антитело (g) может содержать hzVSF_var6, антитело (h) может содержать hzVSF_var7, антитело (i) может содержать hzVSF_var8, антитело (j) может содержать hzVSF_var9, антитело (k) может содержать hzVSF_var10, антитело (l) может содержать hzVSF_var11, антитело (m) может содержать hzVSF_var12, и антитело (n) может содержать hzVSF_var13.
Гуманизированное антитело или фрагмент, связывающиеся с указанным пептидом, могут содержать вариабельную область тяжелой цепи и вариабельную область легкой цепи SEQ ID NO: 10 и SEQ ID NO: 12, SEQ ID NO: 32 и SEQ ID NO: 34, SEQ ID NO: 36 и SEQ ID NO: 38, SEQ ID NO: 40 и SEQ ID NO: 42, SEQ ID NO: 44 и SEQ ID NO: 46, SEQ ID NO: 48 и SEQ ID NO: 50, SEQ ID NO: 52 и SEQ ID NO: 54, SEQ ID NO: 56 и SEQ ID NO: 58, SEQ ID NO: 60 и SEQ ID NO: 62, SEQ ID NO: 64 и SEQ ID NO: 66, SEQ ID NO: 68 и SEQ ID NO: 70, SEQ ID NO: 72 и SEQ ID NO: 74, SEQ ID NO: 76 и SEQ ID NO: 78 или SEQ ID NO: 80 и SEQ ID NO: 82, соответственно, но не ограничены ими.
Конкретно, мышиное антитело может содержать вариабельную область тяжелой цепи, содержащую CDR1 тяжелой цепи SEQ ID NO: 137, CDR2 тяжелой цепи SEQ ID NO: 138 и CDR3 тяжелой цепи SEQ ID NO: 139, и вариабельную область легкой цепи, содержащую CDR1 легкой цепи SEQ ID NO: 134, CDR2 легкой цепи SEQ ID NO: 135 и CDR3 легкой цепи SEQ ID NO: 136, и, конкретнее, содержать вариабельную область тяжелой цепи SEQ ID NO: 9 и вариабельную область легкой цепи SEQ ID NO: 8, но не ограничено ими.
Конкретно, химерное антитело может содержать вариабельную область тяжелой цепи SEQ ID NO: 141 или SEQ ID NO: 142 и вариабельную область легкой цепи SEQ ID NO: 140, и, конкретнее, тяжелую цепь SEQ ID NO: 146 или SEQ ID NO: 148 и легкую цепь SEQ ID NO: 144, но не ограничено ими.
scFv может также включать scFv, полученный для безопасности mVSF, но не ограничен им, и, например, scFv может быть получен с использованием последовательности, показанной на ФИГ. 4. Кроме того, scFv может быть представлен в форме, где вариабельная область тяжелой цепи SEQ ID NO: 131 и вариабельная область легкой цепи SEQ ID NO: 133 связаны линкером. Кроме того, scFv может быть представлен в форме, где вариабельная область тяжелой цепи, кодируемая нуклеотидной последовательностью SEQ ID NO: 130, и вариабельная область легкой цепи, кодируемая нуклеотидной последовательностью SEQ ID NO: 132, связаны линкером. Такой scFv может быть клонирован в вектор экспрессии в Е. coli с нуклеотидной последовательностью SEQ ID NO: 150.
Согласно определенному воплощению настоящего изобретения авторы настоящего изобретения получили гуманизированные антитела, то есть hzVSF_wt, три альтернативных антитела и 13 их вариантов (Пример 5). Кроме того, в результате сравнения иммуногенности полученных гуманизированных антител с имеющимися в продаже лекарственными средствами, утвержденными Управлением по контролю за продуктами и лекарствами (FDA), было подтверждено, что иммуногенность гуманизированных антител была сходна с иммуногенностью лекарственного средства «Хумира», представляющего собой человеческое антитело с наименьшей иммуногенностью среди имеющихся в продаже антител (Таблица 7), посредством чего было подтверждено, что полученные гуманизированные антитела могут быть использованы в качестве безопасных лекарственных средств, квазилекарственных средств, препаратов для накожного применения и так далее без каких-либо неблагоприятных эффектов, которые могут возникать при применении в качестве терапевтических агентов в связи с заболеваниями кожи.
Кроме того, в анализе с Т-клетками было показано, что варианты hzVSF_wt не оказывают существенного влияния на пролиферацию Т-клеток (Таблица 8), что позволило подтвердить низкий риск развития нежелательных реакций, обусловленных действием этих вариантов в качестве иммуногенов, при их применении в клинических исследованиях.
В настоящем изобретении заболевание кожи может представлять собой розовый лишай, опоясывающий герпес, простой хронический лишай, простой герпес, нумуллярную экзему, экзему, контагиозный моллюск, псориаз, простые бородавки, атопический дерматит или аллергический контактный дерматит, и, конкретно, заболевание кожи может представлять собой простой хронический лишай, псориаз, нумуллярную экзему, экзему, атопический дерматит или аллергический контактный дерматит. Тем не менее, заболевание кожи по настоящему изобретению может включать любое заболевание кожи при условии, что оно сопровождается экспрессией VR в ткани кожи или клетках кожи.
В определенном воплощении настоящего изобретения была подтверждена экспрессия пептида SEQ ID NO: 1, имеющего происхождение из виментина, в тканях кожи пациентов с различными заболеваниями кожи, несмотря на отсутствие экспрессии этого пептида в нормальных клетках (ФИГ. 17). Кроме того, было подтверждено, что вещество, специфично связывающееся с пептидом SEQ ID NO: 1, продемонстрировало по существу отличную терапевтическую эффективность в отношении заболеваний кожи, даже у пациентов с простым хроническим лишаем, нумуллярной экземой, экземой, атопическим дерматитом или аллергическим контактным дерматитом (ФИГ. 21-28), и, таким образом, может быть использовано в качестве фармацевтической композиции для предупреждения и лечения различных заболеваний кожи, конкретная этиология которых пока не ясна.
Фармацевтическая композиция может дополнительно содержать фармацевтически приемлемый носитель.
При использовании здесь термин «фармацевтически приемлемый носитель» относится к носителю или разбавителю, не ингибирующему биологические активности или свойства соединения, которое необходимо ввести в организм, не вызывая раздражения у этого организма. Примеры фармацевтически приемлемого носителя, используемого в композиции, которую необходимо изготовить в форме жидкого раствора, включают физиологический раствор, стерильную воду, раствор Рингера, забуференный физиологический раствор, раствор альбумина для инъекций, раствор декстрозы, раствор мальтодекстрина, глицерин, этанол и смесь по меньшей мере одного их компонента, такого как компоненты, подходящие для стерилизации и применения in vivo, и, по необходимости, возможно также добавление другой традиционной добавки (добавок), такой как антиоксидант, буфер, бактериостатический агент и так далее. Кроме того, композиция может быть изготовлена в форме композиций для инъекций, таких как водный раствор, суспензия, эмульсия и так далее, пилюль, капсул, гранул или таблеток посредством дополнительного добавления разбавителя, диспергирующего вещества, поверхностно-активного вещества, связывающего агента, смазывающего агента и так далее.
Фармацевтическая композиция может быть изготовлена в форме различных композиций для перорального или парентерального введения. Для изготовления этих композиций фармацевтическая композиция может быть изготовлена в комбинации с широко применяемым разбавителем или эксципиентом, таким как наполнитель, заполнитель (extender), связывающий агент, увлажнитель, разрыхлитель, поверхностно-активное вещество и так далее. Твердые композиции для перорального введения могут включать таблетки, пилюли, порошки, гранулы, капсулы и так далее, и эти твердые композиции могут быть изготовлены смешиванием с по меньшей мере одним эксципиентом, например, крахмалом, карбонатом кальция, сахарозой или лактозой, желатином и так далее. Помимо простого эксципиента, может быть использован смазывающий агент, такой как стеарат магния, тальк и так далее. Жидкие композиции для перорального введения могут включать суспензии, растворы для перорального введения, эмульсии, сиропы и так далее, и, помимо простого разбавителя, такого как вода или жидкий парафин, в жидкие препараты могут быть включены различные эксципиенты, такие как увлажнители, подсластители, корригенты, консерванты и так далее. Композиции для парентерального введения могут включать стерильные водные растворы, неводные растворители, суспензии, эмульсии, лиофилизированные композиции, суппозитории. Примеры неводных растворителей и суспензий могут включать пропиленгликоль, полиэтиленгликоль, растительные масла, такие как оливковое масло, сложный эфир, подходящий для инъекционного введения, такой как этилолеат, и так далее. Примеры основ для суппозиториев могут включать витепсол, макрогол, Tween 61, масло какао, лаурин (laurinum), глицерожелатин и так далее.
Фармацевтическая композиция может представлять собой композицию любого типа, выбранную из группы, состоящей из таблеток, пилюль, порошков, гранул, капсул, суспензий, растворов для перорального введения, эмульсий, сиропов, стерильных водных растворов, неводных растворителей, суспензий, лиофилизированных композиций и суппозиториев.
Композицию по настоящему изобретению вводят в фармацевтически эффективной дозе.
При использовании здесь термин «фармацевтически эффективная доза» относится к количеству, достаточному для лечения заболеваний с разумным соотношением пользы и риска применительно к лекарственной терапии, и уровень эффективной дозы может быть определен, исходя из факторов, включающих тип субъекта, тяжесть заболевания, возраст, пол, тип заболевания (заболеваний), активность лекарственного средства, чувствительность к лекарственному средству, время введения, путь введения и скорость растворения, продолжительность лечения, факторы, включающие лекарственное(ые) средство (средства), которые необходимо применять одновременно в комбинации, и другие факторы, хорошо известные в области медицины. Композиция по настоящему изобретению может быть введена как отдельный терапевтический агент, в комбинации с другим(и) терапевтическим(и) агентом (агентами) и последовательно или одновременно с традиционным(и) терапевтическим(и) агентом (агентами). Кроме того, композицию по настоящему изобретению можно вводить в однократной дозе или многократных дозах. Важно, чтобы вводимое количество позволяло получить максимальный эффект при минимальном количестве без неблагоприятных эффектов с учетом факторов, описанных выше, и специалист в данной области может легко определить эти факторы. Другой терапевтический агент может представлять собой интерферон, но не ограничен им.
При использовании здесь термин «предупреждение» может относиться ко всем действиям, приводящим к подавлению или задержке начала заболевания посредством введения композиции, и термин «лечение» может относиться ко всем действиям, связанным с облегчением или полезными изменениями симптомов заболевания в результате введения композиции.
Согласно другому аспекту настоящего изобретения предложена косметическая композиция для предупреждения или облегчения заболеваний кожи, содержащая, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1.
Конкретнее, согласно настоящему изобретению предложена косметическая композиция для предупреждения или облегчения заболеваний кожи, содержащая, в качестве активного ингредиента, антитело, специфично связывающееся с пептидом SEQ ID NO: 1, или фрагмент, связывающийся с указанным пептидом.
Вещество, специфично связывающееся с пептидом SEQ ID NO: 1, пептид SEQ ID NO: 1, антитело, фрагмент, связывающийся с указанным пептидом, заболевание кожи и предупреждение соответствуют описанным выше.
В определенном воплощении настоящего изобретения было подтверждено, что вещество, специфично связывающееся с пептидом SEQ ID NO: 1, продемонстрировало по существу отличную терапевтическую эффективность, даже у пациентов с псориазом, простым хроническим лишаем, нумуллярной экземой, экземой, атопическим дерматитом или аллергическим контактным дерматитом (ФИГ. 21-28), и, таким образом, может быть использовано в качестве косметической композиции для предупреждения и облегчения различных заболеваний кожи, конкретная этиология которых пока не ясна.
Косметическая композиция по настоящему изобретению может быть изготовлена в форме композиций, выбранных из группы, состоящей из раствора, мази для местного применения, крема, пены, питательной косметической воды, смягчающей косметической воды, косметической маски, смягчающей воды, латекса, основы для макияжа, духов, мыла, жидкого средства для мытья, пены для ванн, солнцезащитного крема, масла для загара, суспензии, эмульсии, пасты, геля, лосьона, порошка, очищающего средства, содержащего поверхностно-активное вещество, масла, тональной пудры, тональной эмульсии, тонального воска, пластыря и спрея, но не ограничена ими.
Кроме того, косметическая композиция по настоящему изобретению может дополнительно содержать по меньшей мере один косметически приемлемый носитель, смешанный с обычной косметической композицией для кожи. В качестве распространенных ингредиентов для смешивания могут быть использованы, например, масло, вода, поверхностно-активные вещества, увлажняющие средства, низшие спирты, загустители, хелатирующие агенты, красители, консерванты, ароматизаторы и так далее.
Косметически приемлемый носитель, содержащийся в косметической композиции по настоящему изобретению, может различаться, в зависимости от состава композиций.
Когда композиция по настоящему изобретению представляет собой мазь, пасту, крем или гель, в качестве ингредиента-носителя могут быть использованы жидкий животный жир, растительное масло, воск, парафин, крахмал, трагакантовая камедь, производные целлюлозы, полиэтиленгликоль, силикон, бентонит, диоксид кремния, тальк, оксид цинка или их смеси.
Когда композиция по настоящему изобретению представляет собой порошок или спрей, в качестве ингредиента-носителя могут быть использованы лактоза, тальк, диоксид кремния, гидроксид алюминия, силикат кальция, полиамидный порошок или их смеси, и, в частности, когда она представляет собой спрей, в нее может быть дополнительно включен пропеллент, такой как хлорфторуглеводород, пропан/бутан или диметиловый эфир.
Когда композиция по настоящему изобретению представляет собой раствор или эмульсию, в качестве ингредиента-носителя могут быть использованы растворители, солюбилизирующие агенты или эмульгаторы, и, например, могут быть использованы вода, этанол, изопропанол, этилкарбонат, этилацетат, бензиловый спирт, бензилбензоат, пропиленгликоль, 1,3-бутилгликоль, масло. В частности, могут быть использованы хлопковое масло, арахисовое масло, масло из зародышей кукурузы, оливковое масло, касторовое масло и масло семян кунжута, алифатический эфир глицерина, полиэтиленгликоль или алифатический эфир сорбитана.
Когда композиция по настоящему изобретению представляет собой суспензию, в качестве ингредиента-носителя могут быть использованы жидкие разбавители, такие как вода, этанол или пропиленгликоль, суспендирующий агент, такой как этоксилированный изостеариловый спирт, сложный эфир полиоксиэтиленсорбита и сложный эфир полиоксиэтиленсорбитана, микрокристаллическая целлюлоза, метагидроксид алюминия, бентонит, агар, трагакантовая камедь и так далее.
Когда композиция по настоящему изобретению представляет собой мыло, в качестве ингредиента-носителя могут быть использованы соли щелочных металлов и жирных кислот, соли полуэфиров жирных кислот, белковые гидролизаты жирных кислот, изетионат, производные ланолина, алифатические спирты, растительное масло, глицерин, сахара и так далее.
Согласно еще одному аспекту настоящего изобретения предложена квазилекарственная композиция для предупреждения или облегчения заболеваний кожи, содержащая, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1.
Конкретнее, согласно настоящему изобретению предложена квазилекарственная композиция для предупреждения или облегчения заболеваний кожи, содержащая, в качестве активного ингредиента, антитело, специфично связывающееся с пептидом SEQ ID NO: 1, или фрагмент, связывающийся с указанным пептидом.
Вещество, специфично связывающееся с пептидом SEQ ID NO: 1, пептид SEQ ID NO: 1, антитело, фрагмент, связывающийся с указанным пептидом, заболевание кожи и предупреждение соответствуют описанным выше.
Помимо указанных выше компонентов, квазилекарственная композиция по настоящему изобретению может, по необходимости, дополнительно содержать фармацевтически приемлемый носитель, эксципиент или разбавитель. Фармацевтически приемлемый носитель, эксципиент или разбавитель не ограничены при условии сохранения эффекта настоящего изобретения и могут включать, например, наполнители, заполнители, связывающие агенты, увлажняющие агенты, разрыхлители, поверхностно-активные вещества, смазывающие агенты, подсластители, корригенты, консерванты и так далее.
Примеры квазилекарственной композиции по настоящему изобретению могут включать дезинфицирующие очищающие средства, пены для душа, мази, влажные салфетки, агенты для покрытия и так далее, но не ограничены ими. Способ изготовления, доза, способ применения, компоненты и так далее квазилекарственной композиции могут быть подходящим образом выбраны из традиционных методик, известных в данной области.
Согласно еще одному аспекту настоящего изобретения предложен препарат для накожного применения для предупреждения или облегчения заболеваний кожи, содержащий, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1.
Конкретнее, согласно настоящему изобретению предложен препарат для накожного применения для предупреждения или облегчения заболеваний кожи, содержащий, в качестве активного ингредиента, антитело, специфично связывающееся с пептидом SEQ ID NO: 1, или фрагмент, связывающийся с указанным пептидом.
Вещество, специфично связывающееся с пептидом SEQ ID NO: 1, пептид SEQ ID NO: 1, антитело, фрагмент, связывающийся с указанным пептидом, заболевание кожи и предупреждение соответствуют описанным выше.
При использовании здесь термин «препарат для накожного применения» является общим понятием, включающим все вещества, обычно используемые для нанесения на кожу. Неограничивающие примеры композиций, включая фармацевтическую композицию, могут включать пластыри, лосьоны, жидкие мази, жидкости и растворы, аэрозоли, экстракты, мази, жидкие экстракты, эмульсии, суспензии, капсулы, кремы, мягкие или твердые желатиновые капсулы, пластыри или агенты с длительным высвобождением.
Препарат для накожного применения по настоящему изобретению может представлять собой препарат для парентерального введения, изготовленный в твердой, полутвердой или жидкой форме посредством добавления широко используемого неорганического или органического носителя, эксципиента и разбавителя. Для парентерального введения препарат для накожного применения может представлять собой композицию для трансдермального введения, выбранную из группы, состоящей из капель, мазей, лосьонов, гелей, кремов, пластырей, спреев, суспензий и эмульсий, но не ограничен ими.
Носители, эксципиенты и разбавители, которые могут быть включены в препарат для накожного применения, включают лактозу, декстрозу, сахарозу, олигосахариды, сорбит, маннит, ксилит, эритрит, мальтит, крахмал, аравийскую камедь, альгинат, желатин, фосфат кальция, силикат кальция, целлюлозу, метилцеллюлозу, микрокристаллическую целлюлозу, поливинилпирролидон, воду, метилгидроксибензоат, пропилгидроксибензоат, тальк, стеарат магния и минеральное масло.
В случае каждого препарата для накожного применения специалист в данной области может без труда выбрать и включить в смесь другие компоненты, помимо композиции по настоящему изобретению, описанной выше, исходя из состава или цели использования других препаратов для накожного применения, и в этом случае при одновременном нанесении этих компонентов с другими веществами может быть достигнут синергетический эффект.
Согласно еще одному аспекту настоящего изобретения предложен способ предупреждения или лечения заболеваний кожи, включающий введение композиции, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1, субъекту.
Конкретно, согласно настоящему изобретению предложен способ предупреждения или лечения заболеваний кожи, включающий введение композиции, содержащей, в качестве активного ингредиента, антитело, специфично связывающееся с пептидом SEQ ID NO: 1, или фрагмент, связывающийся с указанным пептидом, субъекту.
Способ лечения заболеваний кожи может представлять собой способ, включающий введение фармацевтической композиции, содержащей антитело или дополнительный фармацевтически приемлемый носитель, субъекту с заболеванием кожи или подозрением на такое заболевание. Фармацевтически приемлемый носитель соответствует описанному выше.
Субъект может включать млекопитающих, птиц и так далее, таких как крупный рогатый скот, свиньи, овцы, куры, собаки, люди и так далее, и может включать, без ограничения, любого субъекта, у которого можно лечить заболевания кожи путем введения композиции по настоящему изобретению.
В частности, композиция может быть введена в фармацевтически приемлемом количестве в однократной дозе или многократных дозах. Композиция может быть введена в форме жидкостей, порошков, аэрозолей, капсул, таблеток, покрытых кишечнорастворимой оболочкой, капсул или суппозиториев. Примеры путей введения могут включать внутрибрюшинное, внутривенное, внутримышечное, подкожное, эндотелиальное, пероральное, местное, интраназальное, интрапульмональное, интраректальное введение и так далее, но не ограничены ими. Тем не менее, ввиду расщепления пептидов при их пероральном введении, композиция для перорального введения должна быть изготовлена таким образом, чтобы активный ингредиент был покрыт оболочкой или защищен от разрушения в желудке. Кроме того, фармацевтическая композиция может быть введена с использованием любого устройства, позволяющего обеспечить транспорт активного ингредиента к клетке-мишени.
Согласно еще одному аспекту настоящего изобретения предложено применение композиции для предупреждения или лечения заболеваний кожи, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1.
Согласно еще одному аспекту настоящего изобретения предложено применение композиции, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1, для изготовления лекарственного средства для предупреждения или лечения заболеваний кожи.
Согласно еще одному аспекту настоящего изобретения предложено применение композиции, содержащей, в качестве активного ингредиента, вещество, специфично связывающееся с пептидом SEQ ID NO: 1, для изготовления косметических средств, квазилекарственных средств или препаратов для накожного применения для облегчения заболеваний кожи.
МЕТОД ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
Настоящее изобретение будет описано более подробно посредством Примеров. Тем не менее, эти Примеры приведены исключительно для наглядности и не ограничивают объем настоящего изобретения.
Пример 1: Получение VSF (вирусоподявляющего фактора)
Пример 1-1: Получение chVSF (химерного VSF)
Исходя из того допущения, что основной функциональной частью мышиного VSF (mVSF) является моноклональное антитело, посредством генной инженерии с использованием mVSF и человеческого иммуноглобулина было получено мышиное/человеческое химерное антитело (chAb).
Конкретно, для получения химерного антитела константные области легкой и тяжелой цепей mVSF были заменены константными областями антитела, являющегося человеческим иммуноглобулином (κ, γ2 или γ4). Для chVSF получали вектор экспрессии, используя вектор pCAGGS в качестве матрицы (ФИГ. 1). Вариабельную область тяжелой цепи mVSF (mVH) (SEQ ID NO: 9), включая сайты рестрикции SacI и KpnI, амплифицировали посредством полимеразной цепной реакции (ПЦР). Вариабельную область легкой цепи (mVL) (SEQ ID NO: 8), включая сайты рестрикции ClaI и XhoI амплифицировали посредством ПЦР. Праймеры, использованные при ПЦР, описаны в Таблице 1, и ПЦР проводили на протяжении в общей сложности 35 циклов, включавших 94°С в течение 45 с, 60°С в течение 45 с и 72°С в течение 45 с, и затем при 72°С в течение 10 мин.


Человеческую тяжелую цепь (SEQ ID NO: 11) клонировали с использованием сайтов рестрикции KpnI и SphI, и легкую цепь (SEQ ID NO: 13) клонировали с использованием сайтов рестрикции XhoI и BglI. Для одновременной экспрессии тяжелой цепи и легкой цепи между легкой цепью и тяжелой цепью клонировали внутренний сайт посадки рибосом (IRES), используя сайты рестрикции SphI и ClaI. В сайт рестрикции SalI вводили селектируемый маркер. Соответственно, chVSF получали, как показано на схематической диаграмме, представленной на ФИГ 2.
Пример 1-2: Экспрессия chVSF с использованием двухвекторной системы экспрессии
Клетки HEK 293Т трансфицировали 15 мкг pCAGGS-GFP с использованием 1 мг/мл полиэтиленимина (PEI) для оценки уровней трансфекции и экспрессии. Таким же образом chVSF, полученным в Примере 1-1, трансфицировали клетки HEK 293Т, и через 6 часов среду заменяли средой, содержащей 2% фетальную бычью сыворотку (FBS). Супернатант клеточной культуры собирали каждые 3 суток и присутствовавшие в нем примеси удаляли, используя фильтр (0,45 мкм). chVSF очищали с использованием гранул nProtein A Sepharose®. chVSF элюировали буфером с 0,2 М глицином/HCl (рН 2,5), и в качестве нейтрализующего буфера использовали буфер с 1 М трис-Cl (рН 9,0). Конкретно, после гомогенизации смолы 10-кратным объемом буфера с 1 М трис-Cl (рН 8,0) относительно объема смолы через колонку пропускали культуральный супернатант с VSF. После этого колонку промывали, внося в нее по меньшей мере 5-кратный объем буфера с 0,1 М трис-Cl (рН 8,0) относительно объема колонки. Затем проводили элюирование, внося в колонку 5-кратный объем буфера с 0,2 М глицином/HCl (рН 2,5) относительно объема смолы, получая посредством этого очищенные VSF в пробирке, в которую предварительно был добавлен нейтрализующий буфер. Очищенные VSF затем подтверждали посредством SDS-PAGE.
В результате, как показано на ФИГ. 3, было подтверждено, что chVSF имеет структуру, состоящую из тяжелой цепи (50 кДа) и легкой цепи (25 кДа), имеющих характеристики иммуноглобулина.
Пример 2: Получение одноцепочечного вариабельного фрагмента (scFv)
Одноцепочечный вариабельный фрагмент (scFv) получали, используя вариабельные области VSF. scFv имеет ДНК-последовательность SEQ ID NO: 150, и scFv получали клонированием этой ДНК в рЕТ-22b (+), вектор экспрессии в Е. coli (ФИГ. 4 и 5).
Конкретно, scFV получали связыванием VH и VL mVSF через линкер, встроенный в бактериальный вектор экспрессии рЕТ-22b (+), обработанный изопропилтиогалактозидом (IPTG) для индукции его экспрессии, и затем очищали с использованием Ni-NTA-колонки (ФИГ. 6).
Пример 3: Получение гуманизированного антитела VSF
Гуманизированное антитело hzVSF получали с использованием chVSF на основе Примера 1.
В частности, использовали вектор pdCMV-dhfr, соответствующий двухгенному вектору экспрессии, то есть системе экспрессии, используемой для экспрессии двух разных типов рекомбинантных белков с использованием эукариотической клетки (ФИГ. 7). Этот вектор состоит из двух разных транскрипционных единиц для двух разных типов генов в одном векторе и, благодаря этому, позволяет экспрессировать два разных гена с использованием промотора и сигнальной последовательности полиаденилирования (polyA) в каждой транскрипционной единице, и он представляет собой векторную систему, в которой использован цитомегаловирусный (CMV) промотор, сильный промотор для клеток млекопитающих. С использованием этого промотора получали hzVSF, как показано на ФИГ. 8.
В связи с этим, аминокислотная последовательность вариабельной области тяжелой цепи hzVSF была обозначена SEQ ID NO: 10, и аминокислотная последовательность тяжелой цепи была обозначена SEQ ID NO: 11, в то время как аминокислотная последовательность вариабельной области легкой цепи была обозначена SEQ ID NO: 12, и аминокислотная последовательность легкой цепи была обозначена SEQ ID NO: 13.
Клетки НЕК 293Т трансфицировали 15 мкг pCAGGS-GFP с использованием 1 мг/мл полиэтиленимина (PEI) для оценки уровней трансфекции и экспрессии. Таким же образом chVSF и hzVSF трансфицировали в клетки HEK 293Т, и через 6 часов среду заменяли средой, содержащей 2% FBS. Супернатант клеточной культуры собирали каждые 3 суток и присутствовавшие в нем примеси удаляли, используя фильтр (0,45 мкм). chVSF и hzVSF очищали с использованием гранул nProtein A Sepharose®. chVSF и hzVSF элюировали буфером с 0,2 М глицином/HCl (рН 2,5), и в качестве нейтрализущего буфера использовали буфер с 1 М трис-Cl (рН 9,0). Конкретно, после гомогенизации смолы 10-кратным объемом буфера с 1 М трис-Cl (рН 8,0) относительно объема смолы через колонку пропускали культуральный супернатант с VSF. После этого колонку промывали, внося в нее по меньшей мере 5-кратный объем буфера с 0,1 М трис-Cl (рН 8,0) относительно объема колонки. Затем проводили элюирование, внося в колонку 5-кратный объем буфера с 0,2 М глицином/HCl (рН 2,5) относительно объема смолы, получая посредством этого очищенные VSF в пробирке, в которую был предварительно добавлен нейтрализующий буфер. Очищенные VSF затем подтверждали посредством SDS-PAGE, а их активность подтверждали MVIT-анализом.
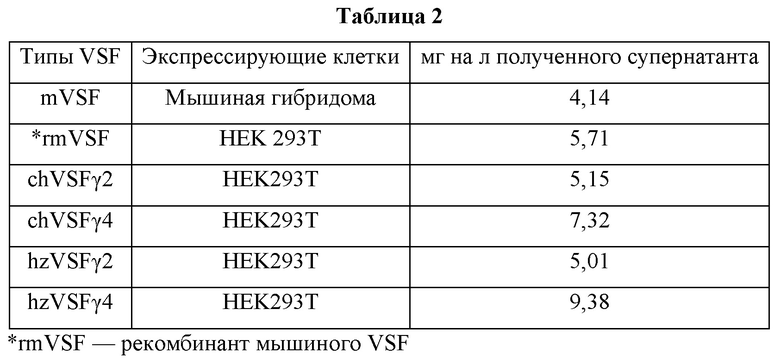
VSF, использованные в эксперименте, показаны в Таблице 2 ниже.
В результате, как видно на ФИГ. 9, было подтверждено, что chVSFγ2, chVSFγ4, hzVSFγ2 и hzVSFγ4 состоят из тяжелой цепи (50 кДа) и легкой цепи (25 кДа), соответственно, имеющих характеристики иммуноглобулина.
Пример 4: Подтверждение физических свойств гуманизированного антитела VSF
Физические свойства hzVSF, полученного в Примере 3, подтверждали следующим образом.
Пример 4-1: Подтверждение основных паттернов молекулярной массы и чистоты
Паттерны молекулярной массы и чистоту подтверждали посредством SDS-PAGE в восстанавливающих и невосстанавливающих условиях. Конкретно, hzVSF_v13 окрашивали красителем Кумасси с применением SDS-PAGE в соответствии с молекулярной массой, посредством чего молекулярная масса и чистота были подтверждены.
В результате, как показано на ФИГ. 10, на дорожке 1 невосстанавливающего геля основную полосу наблюдали в положении, в котором следовало ожидать появления антитела IgG (150 кДа), и на дорожке 2 восстанавливающего геля наблюдали полосы, соответствующие положениям тяжелой цепи (приблизительно 50 кДа) и легкой цепи (приблизительно 25 кДа) иммуноглобулина G (IgG), что подтверждает таким образом, что hzVSF_v13 демонстрировал общий паттерн антитела IgG.
Пример 4-2: Подтверждение молекулярной массы, паттерна гликозилирования, вариабельности размера и так далее
Для подтверждения молекулярной массы, паттерна гликозилирования, вариабельности размера hzVSF_v13 и так далее проводили жидкостную хроматографию/масс-спектрометрию. Проводили HPLC небольшого количества hzVSF_v13 и наблюдали полученные пики.
В результате, было подтверждено, что hzVSF_v13 демонстрировал характеристики IgG (ФИГ. 11). При анализе интактной массы (intact mass) наблюдали общую молекулярную массу hzVSF_v13 (приблизительно 140 кДа), и отмеченные паттерны пиков соответствовали обычному гликозилированному IgG (например, G0/G0, G0F/G1, G1/G1 и так далее). Кроме того, после дегликозилирования наблюдали тяжелую цепь (приблизительно 49 кДа) и легкую цепь (приблизительно 23 кДа). Исходя из молекулярной массы тяжелой цепи, где гликан был удален обработкой пептид-N-гликозидазой F (PNGase F), и тяжелой цепи без обработки пептид-N-гликозидазой F, удалось подтвердить обычный гликановый паттерн IgG (G0F, G1F и G2F).
Пример 4-3: Подтверждение чистоты и агрегации
Для подтверждения чистоты и агрегации hzVSF_v13 проводили SEC-HPLC.
Условия SEC-HPLC были следующими:
- система HPLC: Dionex Ultimate 3000;
- колонка: Tosoh TSKgel G3000 SWxl;
- подвижная фаза: фосфатный буфер, 0,5 мл/мин;
- вводимый объем: 10 мкл.
В результате, 92,44% основного пика наблюдали в положении, соответствующем мономерам типичного антитела IgG (при времени удерживания приблизительно 16 минут), и приблизительно 6,84% пика наблюдали в положении, соответствующем димерам типичного антитела IgG (при времени удерживания приблизительно 13 минут) (ФИГ. 12).
Пример 4-4: Подтверждение pi и гетерогенности заряда
Для подтверждения изоэлектрической точки hzVSF_v13 проводили электрофорез, используя гель с градиентом от рН 3 до рН 10.
В результате, как показано на ФИГ. 13, было продемонстрировано, что hzVSF_v13 имеет pi 7,7, и, помимо основных полос, были также отмечены кислые/основные изоформы. Это соответствует изомерам, обычно наблюдаемым у антител IgG (например, дезаминирование в С-концевой области).
Приведенные выше результаты подтверждают, что физические свойства гуманизированных антител hzVSF_v13 по настоящему изобретению сходны с физическими свойствами антител IgG.
Пример 5: Получение вариантов hzVSF, представляющих собой гуманизированные антитела со сниженной иммуногенностью
Пример 5-1: Получение альтернативных hzVSF
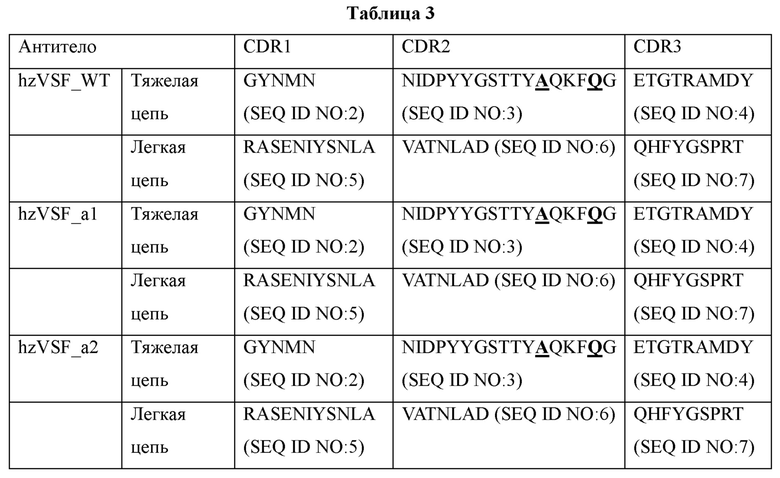
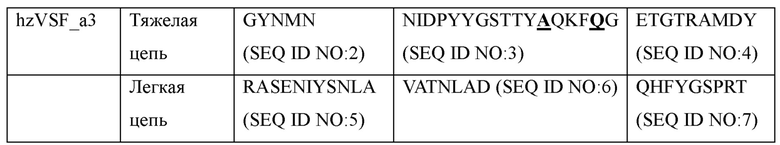
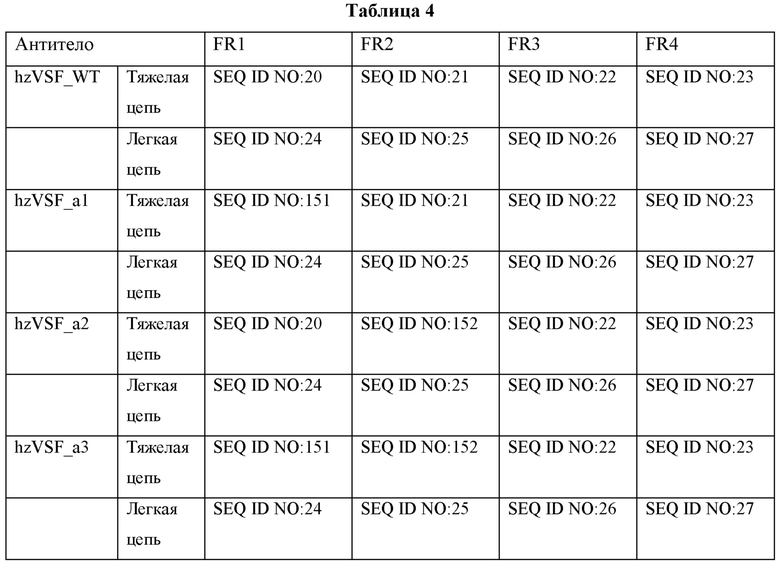
Три альтернативных hzVSF были получены на основе hzVSF, полученного в Примере 3. Активность каждого альтернативного антитела была близка к или ниже активности дикого типа (0,5 =≤ 1 ЕД < 1 мг/мл) (Таблицы 3 и 4). Аминокислотные последовательности CDR1-CDR3 каждого из альтернативных антител показаны в Таблице 3, и аминокислотные последовательности FR1-FR4 каждого из вариантов показаны в Таблице 4.
Пример 5-2: Получение вариантов hzVSF
Варианты hzVSF для фактического применения in vivo были получены на основе hzVSF, полученного в Примере 3, посредством снижения иммуногенности и созревания аффинности. В результате, были получены в общей сложности 13 вариантов (Таблицы 5 и 6). Аминокислотные последовательности CDR1-CDR3 каждого из вариантов показаны в Таблице 5, и аминокислотные последовательности FR1-FR4 каждого из вариантов показаны в Таблице 6.
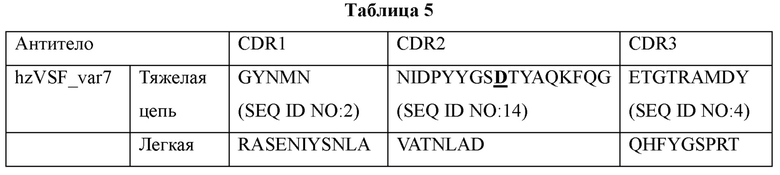
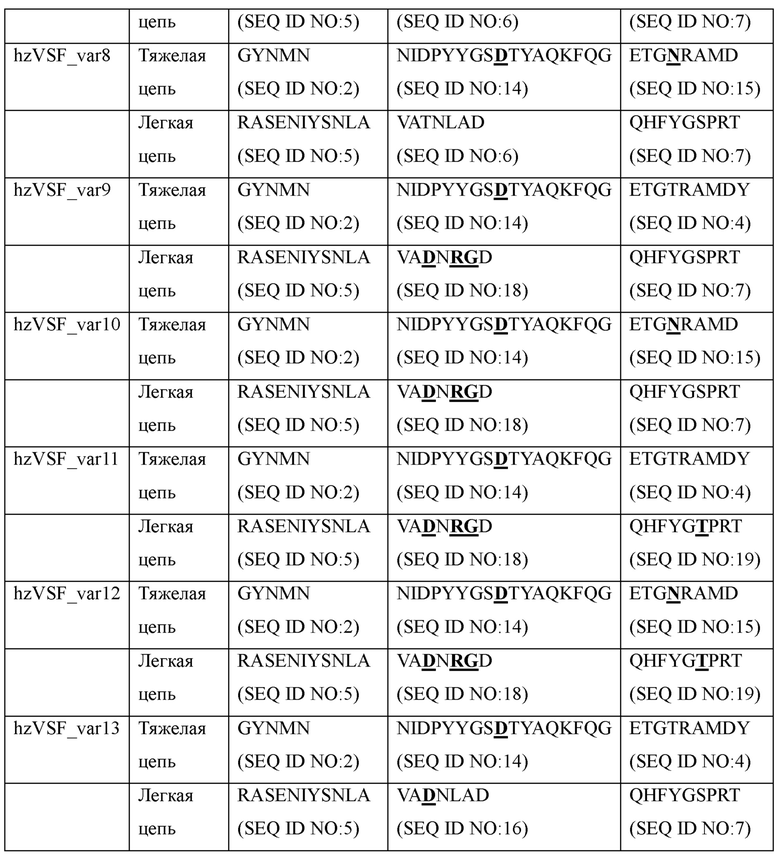
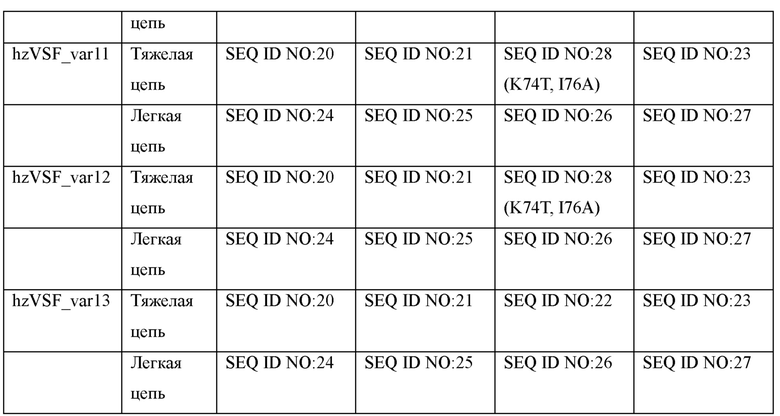
Было подтверждено, что все 13 вариантов, полученные выше, имели сниженную иммуногенность по сравнению с антителами дикого типа.
Пример 5-3: Подтверждение числа эпитопов у hzVSF дикого типа и его вариантов
Было проведено сравнение числа эпитопов у hzVSF дикого типа, hzVSF_var12 и hzVSF_var13, являющихся типичными вариантами с иммуногенностью ниже, чем у дикого типа; и четырех типов активно продаваемых антител, являющихся лекарственными средствами, представленными в настоящее время на фармацевтическом рынке. В результате, была подтверждена возможность относительной иммуногенности при каждом HLA класса II, как показано в Таблице 7.
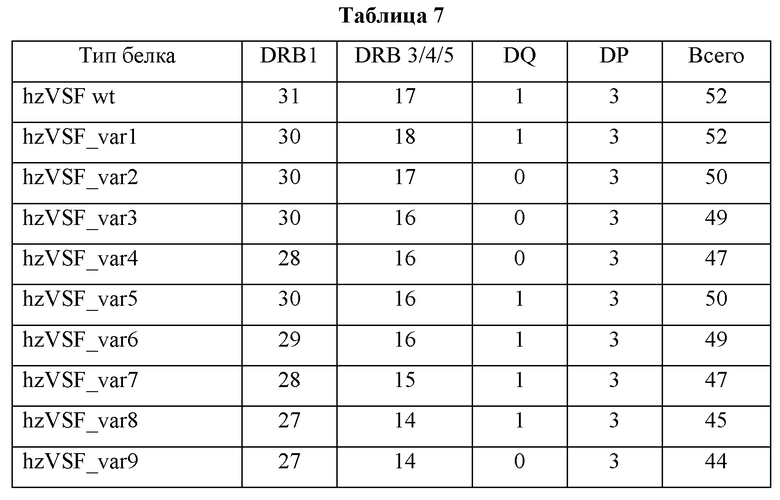
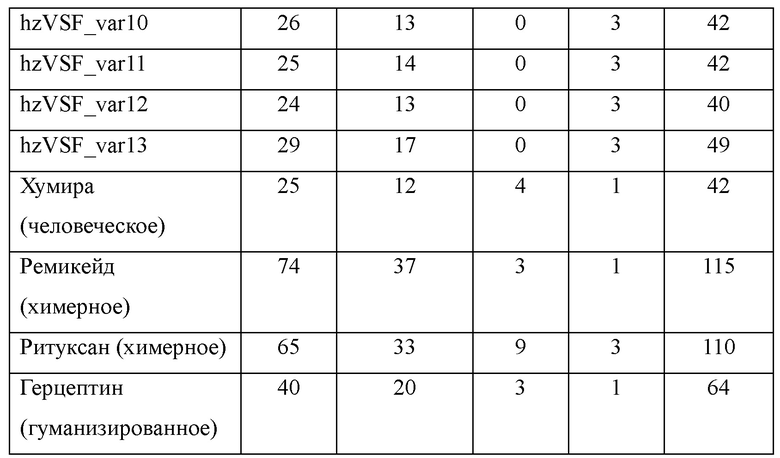
В Таблице 7 более высокий общий показатель означает более высокую вероятность неблагоприятных эффектов, обусловленных HLA класса II. Исходя из представленных выше результатов, было подтверждено, что число эпитопов у hzVSF_var12 и hzVSF_var13 со сниженной иммуногенностью по настоящему изобретению было сходно с числом эпитопов у лекарственного средства «Хумира», имеющего наименьшее число эпитопов среди четырех рассматриваемых типов лекарственных средств. Эти результаты показывают, что гуманизированные антитела по настоящему изобретению будут иметь очень низкий уровень возможных серьезных побочных эффектов, и, таким образом, подтверждают безопасность гуманизированных антител по настоящему изобретению в качестве лекарственных средств.
Пример 5-4: Подтверждение Т-клеточного анализа вариантов hzVSF
Для оценки иммуногенности hzVSF_var12 и hzVSF_var13 было проведено подтверждение возможного эффекта вещества, изученного с применением Т-клеточного анализа Lonza in vitro, на пролиферацию Т-клеток из образцов крови 51 здорового донора. Неразделенные мононуклеарные клетки периферической крови (РВМС) каждого донора обрабатывали hzVSF_var12, hzVSF_var13 или гемоцианином фиссуреллы (KLH) и культивировали на протяжении 7 суток. KLH представляет собой кислородсодержащий металлопротеин, который можно использовать в качестве белка-носителя при получении антител и который был использован в качестве положительного контроля ввиду его способности эффективно вызывать иммунные ответы. Затем измеряли долю (%) Т-клеток, окрашенных CD3+CD4+Edu+. В результате, KLH продемонстрировал пролиферацию Т-клеток у 45 доноров крови (частота ответа 88%), в то время как hzVSF_var12 и hzVSF_var13 индуцировали пролиферацию Т-клеток только у 3 субъектов в ответ на указанное вещество (частота ответа 5,8%) (ФИГ. 14).
В то же время, с использованием РВМС, культивированных в качестве контроля на протяжении 7 суток, был рассчитан индекс стимуляции (SI) пролиферации Т-клеток, индуцированной KLH, hzVSF_v12 или hzVSF_v13. При значении SI более 2 следует ожидать высокой иммуногенности при применении у людей. При значении SI менее 0,5, наоборот, следует ожидать ингибирования пролиферации Т-клеток. В результате, было подтверждено, что hzVSF_v12 и hzVSF_v13 показали низкие значения SI, составившие 1,12 и 1,03, соответственно, входившие в диапазон от 0,6 до 2 включительно. В отличие от этого, KLH, использованный в качестве положительного контроля, показал высокое значение SI, составившее 3,91 (ФИГ. 15 и 16; и Таблица 8).
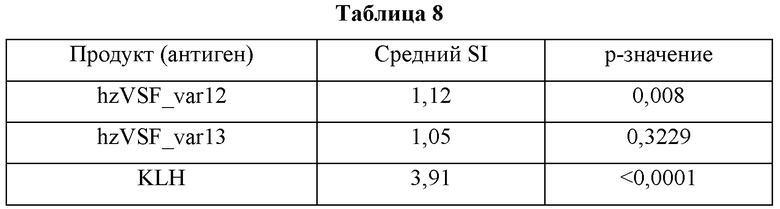
Исходя из представленных выше результатов, следует ожидать, что hzVSF_v12 и hzVSF_v13 будут иметь меньшую иммуногенность при введении пациентам с заболеваниями кожи и, следовательно, меньше побочных эффектов.
Пример 6: Изучение взаимодействия нормальных человеческих тканей и VSF
Далее описаны различные подтверждения терапевтических эффектов hzVSF (далее - VSF), полученного и подтвержденного в Примерах 1-5, на заболевания кожи.
Сначала для изучения взаимодействия нормальных человеческих тканей и VSF было проведено иммуногистохимическое окрашивание с использованием mVSF и тканевой матрицы (tissue microarray, ТМА), микропрепарата, на котором расположены нормальные ткани основных органов человека.
В результате, никакого окрашивания нормальных тканей mVSF отмечено не было (ФИГ. 17). Таким образом, видно, что VR, являющийся рецептором VSF, не экспрессирован в нормальных тканях, и VSF будет действовать на клетки кожи только при заболеваниях кожи. Кроме того, VSF не влияет на нормальные ткани и, таким образом, может оказывать отличные фармакологические эффекты при незначительных побочных эффектах, даже при использовании в виде фармацевтической композиции и так далее.
Пример 7: Подтверждение экспрессии VR при различных заболеваниях кожи
Поскольку в Примере 6 было подтверждено, что VR не экспрессирован в нормальных тканях (нормальных клетках), было проведено подтверждение экспрессии VR в тканях кожи при заболеваниях кожи.
Сначала было проведено иммуногистохимическое окрашивание с использованием mVSF и микропрепаратов, на которых были зафиксированы ткани кожи пациентов с различными заболеваниями кожи. На каждом этапе конкретная экспериментальная методика была следующей.
Пример 7-1: Заключение в парафин
Образцы кожи, полученные от пациентов с заболеванием кожи, фиксировали на протяжении 48 часов в 10%-м нейтральном забуференном формалине (NBF). Фиксированные образцы кожи извлекали из фиксирующего раствора (10%-й NBF) и промывали IX фосфатно-солевым буферным раствором (PBS) на протяжении 30 минут, используя мешалку. После удаления IX PBS проводили обезвоживание в мешалке с использованием 70%-го, 90%-го и 100%-го спирта, в каждом случае на протяжении 1 часа. Затем проводили просветление в виде предварительной обработки раствором ксилола и 100%-го спирта (1:1) на протяжении 1 часа. После этого просветление проводили с использованием ксилола 3 раза за 1 час с последующей пропиткой с использованием парафина, расплавленного при 60°С 2 раза за 1 час. Затем пропитку проводили посредством обработки образцов кожи парафином, расплавленным в термостате при 60°С, на протяжении 12 часов. 50% парафина, расплавленного при 60°С, заливали в форму с использованием аппарата для приготовления парафиновых срезов и затем добавляли в нее еще 50% расплавленного парафина. Затем парафиновую форму сушили на протяжении 1 часа на холоде и 6 часов на экспериментальном столе.
После сушки форму удаляли, получая парафиновый блок, и охлаждали его (4°С). Конкретно, 80% холодильной емкости заполняли льдом и добавляли подходящее количество водопроводной воды для поддержания холода, затем охлажденный парафиновый блок помещали в холодильную емкость в направлении образца. Это связано с возможностью разрыва тканей при хранении блока в неохлажденном состоянии, вследствие чего получение тканей надлежащего качества может оказаться невозможным.
Затем ткани нарезали с шагом 4 мкм, используя микротом, полученные срезы погружали в 50%-й спирт и затем помещали на баню с постоянной температурой 43°С. Полученные срезы тканей кожи фиксировали на предметных стеклах. Полученные микропрепараты помещали на нагреватель, разогретый до 43°С, сушили в течение 1 часа и затем охлаждали (4°С).
Пример 7-2: Окрашивание гематоксилином-эозином (Н&Е-окрашивание)
Микропрепараты с образцами кожи оставляли в ксилоле на 10 минут. Ксилол заменяли на новый и оставляли микропрепараты в нем на 10 минут, после чего оставляли их в ксилоле со 100%-м спиртом (1:1) на 3 минуты. Затем проводили депарафинизацию, оставляя микропрепараты в 100%-м, 95%-м, 90%-м, 70%-м и 50%-м спирте, по 3 минуты в каждом. После гидратации микропрепаратов на протяжении 10 минут в потоке водопроводной воды их оставляли в гематоксилине Харриса на 3 минуты для первоначального окрашивания ядер. Затем на протяжении 5 минут проводили промывку в потоке водопроводной воды.
Микропрепараты обесцвечивали, добавляя 1%-ю соляную кислоту (несколько капель), для удаления гематоксилинового красителя и оставляли в эозине на 3 минуты для окрашивания окружающих тканей с последующей промывкой продолжительностью 3 минуты в потоке водопроводной воды. После этого микропрепараты оставляли в 70%-м, 90%-м, 95%-м и 100%-м спирте, по 3 минуты в каждом, и затем оставляли их в ксилоле на 5 минут и заключали в органический растворитель. Микропрепараты сушили в вытяжном шкафу до исчезновения запаха ксилола и изучали ткани под микроскопом.
Пример 7-3: Иммуногистохимическое (ИГХ) окрашивание
Микропрепараты с образцами кожи оставляли в ксилоле на 10 минут. Ксилол заменяли на новый и оставляли микропрепараты в нем на 10 минут, после чего оставляли их в ксилоле со 100%-м спиртом (1:1) на 3 минуты. Затем проводили депарафинизацию, оставляя микропрепараты в 100%-м, 95%-м, 90%-м, 70%-м и 50%-м спирте, по 3 минуты в каждом. После гидратации микропрепаратов на протяжении 10 минут в потоке водопроводной воды их оставляли в 3%-й перекиси водорода на 5 минут и промывали в PBS два раза по 3 минуты. Микропрепараты помещали в демаскирующий буфер и кипятили в микроволновой печи (95°С) на протяжении 10 минут (добавляя буфер по мере его выкипания во избежание высыхания тканей). Буфер охлаждали при комнатной температуре на протяжении 30 минут и проводили промывку в PBS, два раза по 3 минуты. 1%-й бычий сывороточный альбумин (BSA) получали с использованием 1×TBST (50 мМ трис, 150 мМ NaCl, 0,1% Tween 20) и оставляли стоять при комнатной температуре на 1 час. Микропрепараты инкубировали с mVSF (1:50) в течение ночи при 4°С в камере с постоянной влажностью и промывали Зраза по 10 минут, используя 1×TBST. Затем микропрепараты инкубировали с антителами против мышиного IgG, конъюгированными с пероксидазой хрена (HRP) (1:2000), на протяжении 30 минут при комнатной температуре и промывали 3 раза по 10 минут, используя 1×TBST. Затем микропрепараты оставляли в растворе диаминобензидина (DAB) и перекиси водорода (1:20) на 5 минут для выявления хромогенов, после чего оставляли их в гематоксилине Харриса на 30 секунд для окрашивания ядер с последующей промывкой в потоке водопроводной воды. В завершение, микропрепараты заключали в водорастворимую среду.
Пример 7-4: Подтверждение результата по экспрессии VR в тканях кожи пациентов с заболеваниями кожи
Таким же образом, как описано выше, экспрессия VR, рецептора VSF в тканях кожи пациентов с различными заболеваниями кожи и биомаркера, экспрессируемого в клетках кожи только при заболеваниях кожи, была подтверждена посредством иммуногистохимического окрашивания.
Конкретно, у пациентов с заболеваниями кожи проводили пункционную биопсию ткани кожи для получения парафиновых блоков описанным выше методом. Соответственно, для подтверждения экспрессии VR проводили Н&Е-окрашивание и иммуногистохимическое окрашивание с использованием mVSF.
В результате, как показано на ФИГ. 18-20, VR был экспрессирован во всех тканях кожи 10 пациентов с розовым лишаем и в 90% или более в тканях кожи пациентов с опоясывающим герпесом, простым хроническим лишаем и простым герпесом. Кроме того, VR был экспрессирован в 67% или более в тканях кожи пациентов с нумуллярной экземой, контагиозным моллюском, псориазом, простыми бородавками, атопическим дерматитом и аллергическим контактным дерматитом. В заключение, экспрессия VR была подтверждена в образцах ткани кожи 88 пациентов с заболеваниями кожи (частота ответа 80%) из 110 пациентов.
Таким образом, VR, который может связываться с VSF, экспрессирован в тканях кожи пациентов с розовым лишаем, простым хроническим лишаем, нумуллярной экземой, псориазом, атопическим дерматитом и так далее, которые являются заболеваниями кожи, точная этиология которых пока не ясна. Поэтому это может означать, что VSF будет оказывать лечебный эффект при заболеваниях кожи.
Соответственно, терапевтический эффект VSF у пациентов с заболеваниями кожи был изучен более подробно.
Пример 8: Оценка терапевтической эффективности VSF у пациентов с заболеваниями кожи
Целью данного Примера было определение терапевтического эффекта VSF у пациентов с заболеваниями кожи.
Для более объективной оценки эффективности это исследование проводили под руководством дерматологов у пациентов, которые добровольно попросили применить hzVSF.
Эффективность hzVSF оценивали с использованием 50 мкл 5 мг/мл hzVSF, растворенного в PBS, для каждого нанесения, и эффект лечения оценивался дерматологами визуально.
Пример 8-1: Подтверждение терапевтической эффективности VSF у пациентов с псориазом
У пациента 1 с псориазом, у которого был псориаз правой ноги, hzVSF был нанесен на очаг в области колена при первом визите (17 августа 2016 г.) и, дополнительно, 29 августа 2016 г. и 7 сентября 2016 г.
В результате, при осмотре очага в области колена 15 сентября 2016 г. в месте, где был нанесен hzVSF, был отмечен существенный терапевтический эффект по сравнению с другими областями, и при повторном осмотре области очага 20 октября 2016 г., примерно через месяц после четвертого нанесения, проведенного 15 сентября 2016 г., было подтверждено, что нанесение уменьшило область очага на 90% или более (ФИГ. 21).
Таким образом, видно, что hzVSF, полученный в Примере 5, оказывает отличный терапевтический эффект на заболевания кожи, сопровождающиеся экспрессией VR, такие как псориаз.
В то же время, у пациента 2 с псориазом, у которого был псориаз спины, при первом визите (19 августа 2016 г.) была проведена биопсия для подтверждения экспрессии VR, рецептора hzVSF, иммуногистохимическим окрашиванием, которая позволила подтвердить степень экспрессии VR. При первом визите hzVSF был нанесен на очаг на спине, и при осмотре области очага через пять дней, 24 августа, степень образования кератина и покраснения была снижена на 50% или более (ФИГ. 22). Таким образом, отличный эффект лечения был подтвержден и у пациента 2.
Пример 8-2: Подтверждение терапевтической эффективности VSF у пациентов с простым хроническим лишаем
У пациента 1 с простым хроническим лишаем был очаг на правой ноге. С первого визита (7 августа 2016 г.) hzVSF наносили на область очага на голени один раз в 5 дней. При осмотре области очага 22 августа 2016 г. после третьего нанесения было подтверждено подавление образования кератина и покраснения на 70% или более (ФИГ. 23).
После этого hzVS также наносили на область очага на лодыжке. Конкретно, hzVS наносили на область голени в общей сложности шесть раз до 2 сентября 2016 г. и проводили осмотр через месяц, 2 октября 2016 г. В результате, было подтверждено сохранение терапевтического эффекта без рецидивов заболевания. Лодыжку обрабатывали hzVS в общей сложности три раза до 2 сентября 2016 г. и проводили осмотр через месяц, 2 октября 2016 г. В результате, было подтверждено уменьшение области очага на 50% или более (ФИГ. 23).
У пациента 2 с простым хроническим лишаем были очаги на лодыжках обеих ног, и hzVSF наносили на них с первого визита (7 августа 2016 г.). При осмотре областей очагов 30 сентября 2016 г., примерно через шесть недель после второго нанесения, проведенного 19 августа 2016 г., был продемонстрирован терапевтический эффект с почти полным излечением очагов. В частности, после нанесения hzVSF область образования кератина почти исчезла, и был продемонстрирован терапевтический эффект с уменьшением области очага на 90% или более (ФИГ. 24).
У пациента 3 с простым хроническим лишаем были очаги на голени правой ноги, и hzVSF был впервые нанесен при первом визите (17 сентября 2016 г.). При осмотре очага 19 сентября 2016 г., через два дня, было подтверждено уменьшение очага на 50% или более, что позволило подтвердить отличный терапевтический эффект hzVSF (ФИГ. 25).
Пример 8-3: Подтверждение терапевтической эффективности VSF у пациентов с нумуллярной экземой
У пациента с нумуллярной экземой были очаги на правой икре, и нанесение hzVSF проводили два раза в сутки, начиная с первого визита (9 февраля 2018 г.). При осмотре области поражения 23 февраля 2018 г. и 26 марта 2018 г. было визуально подтверждено улучшение в области очага, и значение индекса EASI, позволяющего оценить эритему, отек/бугорки/папулы, осаднение и лихенификацию, на третьем визите (через 45 суток) было снижено, по сравнению с первым визитом, с 11 до 4. Кроме того, на третьем визите был также снижен показатель зуда, с 10 до 5, что позволило подтвердить терапевтический эффект (ФИГ. 26).
Пример 8-4: Количественное измерение терапевтической эффективности VSF у пациентов с заболеваниями кожи
hzVSF наносили два раза в сутки на области очагов у пациентов с простым хроническим лишаем, нумуллярной экземой, экземой, атопическим дерматитом и псориазом, начиная с первого визита, и проводили осмотр областей очагов на втором и третьем визитах. В случае простого хронического лишая средняя продолжительность эксперимента, от первого визита до третьего визита, составила 40 суток, и значение индекса EASI было снижено, в среднем, с 8,5 до 4, и показатель зуда- с 6,5 до 2,5. В случае нумуллярной экземы средняя продолжительность эксперимента, от первого визита до третьего визита, составила 36 суток, и значение индекса EASI было снижено, в среднем, с 8,3 до 3, и показатель зуда- с 8 до 3. В случае экземы продолжительность эксперимента, от первого визита до третьего визита, составила 35 суток, и значение индекса EASI было снижено с 5 до 1, и показатель зуда - с 5 до 2. В случае атопического дерматита продолжительность эксперимента, от первого визита до третьего визита, составила 42 суток, и значение индекса EASI было снижено с 10 до 4, и показатель зуда - с 6 до 2. В случае дерматита продолжительность эксперимента, от первого визита до третьего визита, составила 35 суток, и значение индекса EASI было снижено с 6 до 3, и показатель зуда - с 5 до 4. В случае псориаза средняя продолжительность эксперимента, от первого визита до третьего визита, составила 26 суток, и значение индекса PASI было снижено, в среднем, с 8 до 3,6, и показатель зуда- с 6,3 до 2,6. Исходя из этих результатов, было подтверждено визуальное улучшение в областях очагов и снижение значений индекса EASI, позволяющего оценить эритему, отек/бугорки/папулы, осаднение и лихенификацию, и индекса PASI, позволяющего оценить эритему, утолщение и шелушение кожи, на третьем визите, по сравнению с первым визитом, при всех заболеваниях кожи. Кроме того, было также подтверждено уменьшение зуда на третьем визите, что позволило подтвердить терапевтический эффект (ФИГ. 27 и 28).
В заключение, VSF по настоящему изобретению, то есть hzVSF, полученный в Примерах 5 и 8, может демонстрировать отличный терапевтический эффект при нанесении на кожу при заболеваниях, сопровождающихся экспрессией VR, таких как псориаз, простой хронический лишай, розовый лишай, простой герпес, нумуллярная экзема, контагиозный моллюск, простые бородавки, атопический дерматит и аллергический контактный дерматит. Таким образом, можно предположить, что VSF может быть эффективно использован в качестве терапевтической композиции для предупреждения и лечения заболеваний кожи, сопровождающихся экспрессией VR, и так далее.
Специалисту в данной области будет ясно, что настоящее изобретение может быть воплощено в других конкретных формах без выхода за рамки его сущности или основных признаков. Описанные воплощения следует рассматривать во всех отношениях только как наглядные и неограничивающие. Таким образом, объем настоящего изобретения определен приложенной формулой изобретения, но не предшествующим описанием. Все изменения в рамках значения и диапазона эквивалентности формулы изобретения следует рассматривать как включенные в объем настоящего изобретения.
--->
Список последовательностей
<110> IMMUNEMED INC.
<120> Composition for prevention or treatment of skin disease
comprising a matter specifically binding to peptide derived from
vimentin
<130> OPA18283-RU
<150> 10-2017-0110924
<151> 2017-08-31
<160> 162
<170> KopatentIn 2.0
<210> 1
<211> 153
<212> PRT
<213> Artificial Sequence
<220>
<223> Выделенный фрагмент виментина (142-294)
<400> 1
Gly Lys Ser Arg Leu Gly Asp Leu Tyr Glu Glu Glu Met Arg Glu Leu
1 5 10 15
Arg Arg Gln Val Asp Gln Leu Thr Asn Asp Lys Ala Arg Val Glu Val
20 25 30
Glu Arg Asp Asn Leu Ala Glu Asp Ile Met Arg Leu Arg Glu Lys Leu
35 40 45
Gln Glu Glu Met Leu Gln Arg Glu Glu Ala Glu Asn Thr Leu Gln Ser
50 55 60
Phe Arg Gln Asp Val Asp Asn Ala Ser Leu Ala Arg Leu Asp Leu Glu
65 70 75 80
Arg Lys Val Glu Ser Leu Gln Glu Glu Ile Ala Phe Leu Lys Lys Leu
85 90 95
His Glu Glu Glu Ile Gln Glu Leu Gln Ala Gln Ile Gln Glu Gln His
100 105 110
Val Gln Ile Asp Val Asp Val Ser Lys Pro Asp Leu Thr Ala Ala Leu
115 120 125
Arg Asp Val Arg Gln Gln Tyr Glu Ser Val Ala Ala Lys Asn Leu Gln
130 135 140
Glu Ala Glu Glu Trp Tyr Lys Ser Lys
145 150
<210> 2
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR1 тяжелой цепи
<400> 2
Gly Tyr Asn Met Asn
1 5
<210> 3
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR2 тяжелой цепи
<400> 3
Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 4
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR3 тяжелой цепи
<400> 4
Glu Thr Gly Thr Arg Ala Met Asp Tyr
1 5
<210> 5
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR1 легкой цепи
<400> 5
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala
1 5 10
<210> 6
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR2 легкой цепи
<400> 6
Val Ala Thr Asn Leu Ala Asp
1 5
<210> 7
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR3 легкой цепи
<400> 7
Gln His Phe Tyr Gly Ser Pro Arg Thr
1 5
<210> 8
<211> 169
<212> PRT
<213> Artificial Sequence
<220>
<223> mVSF_VL
<400> 8
Met Ser Val Pro Thr Gln Val Leu Gly Leu Leu Leu Leu Trp Leu Thr
1 5 10 15
Gly Asn Glu Asp Ser Thr Arg Asn Leu Leu Ser Phe Leu His Ser Val
20 25 30
Leu Leu Gly Leu Asn Glu Asn Ser Leu Val Arg Glu Leu Ile Met Trp
35 40 45
Val Ser Val Phe Asn Phe Pro Ile Val Gly Ala Arg Cys Asp Ile Gln
50 55 60
Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Val Gly Glu Thr Val
65 70 75 80
Thr Met Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp
85 90 95
Tyr Gln Gln Lys Gln Gly Lys Ser Pro Gln Leu Leu Val Tyr Val Ala
100 105 110
Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
115 120 125
Gly Thr Gln Phe Ser Leu Lys Ile Asn Ser Leu Gln Pro Glu Asp Phe
130 135 140
Gly Ser Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg Thr Phe Gly
145 150 155 160
Gly Gly Thr Lys Leu Glu Ile Lys Arg
165
<210> 9
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> mVSF_VH
<400> 9
Glu Ile Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ser Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Lys Gln Ser His Gly Lys Ser Leu Glu Trp Ile
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Asn Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Ser Val Thr Val Ser Ser
115
<210> 10
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_WT
<400> 10
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 11
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_WT - HzVSF hVH.hCgamma4_WT
<400> 11
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 12
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_WT
<400> 12
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 13
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_WT
<400> 13
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 14
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR2 тяжелой цепи
<400> 14
Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 15
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR3 тяжелой цепи
<400> 15
Glu Thr Gly Asn Arg Ala Met Asp
1 5
<210> 16
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR2 легкой цепи
<400> 16
Val Ala Asp Asn Leu Ala Asp
1 5
<210> 17
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR2 легкой цепи
<400> 17
Val Ala Asp Asn Leu Gly Asp
1 5
<210> 18
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR2 легкой цепи
<400> 18
Val Ala Asp Asn Arg Gly Asp
1 5
<210> 19
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR3 легкой цепи
<400> 19
Gln His Phe Tyr Gly Thr Pro Arg Thr
1 5
<210> 20
<211> 30
<212> PRT
<213> Artificial Sequence
<220>
<223> FR1 тяжелой цепи
<400> 20
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr
20 25 30
<210> 21
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> FR2 тяжелой цепи
<400> 21
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met Gly
1 5 10
<210> 22
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> FR3 тяжелой цепи
<400> 22
Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr Met Glu
1 5 10 15
Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg
20 25 30
<210> 23
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> FR4 тяжелой цепи
<400> 23
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 24
<211> 23
<212> PRT
<213> Artificial Sequence
<220>
<223> FR1 легкой цепи
<400> 24
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys
20
<210> 25
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> FR2 легкой цепи
<400> 25
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 26
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> FR3 легкой цепи
<400> 26
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 27
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> FR4 легкой цепи
<400> 27
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 28
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> FR3 тяжелой цепи
<400> 28
Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr Met Glu
1 5 10 15
Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Ala Arg
20 25 30
<210> 29
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_a1_gamma4
<400> 29
Glu Ile Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 30
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_a2_gamma4
<400> 30
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 31
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_a3_gamma4
<400> 31
Glu Ile Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 32
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V1
<400> 32
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 33
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V1
<400> 33
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 34
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V1
<400> 34
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 35
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V1
<400> 35
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 36
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V2
<400> 36
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 37
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V2
<400> 37
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 38
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V2
<400> 38
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Leu Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 39
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V2
<400> 39
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Leu Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 40
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V3
<400> 40
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 41
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V3
<400> 41
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 42
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V3
<400> 42
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 43
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V3
<400> 43
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 44
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V4
<400> 44
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 45
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V4
<400> 45
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 46
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V4
<400> 46
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Thr Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 47
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V4
<400> 47
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Thr Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 48
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V5
<400> 48
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 49
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V5
<400> 49
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 50
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V5
<400> 50
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 51
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V5
<400> 51
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 52
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V6
<400> 52
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 53
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V6
<400> 53
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 54
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V6
<400> 54
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 55
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V6
<400> 55
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 56
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V7
<400> 56
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 57
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V7
<400> 57
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 58
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V7
<400> 58
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 59
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V7
<400> 59
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 60
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V8
<400> 60
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Asn Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 61
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V8
<400> 61
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Asn Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 62
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V8
<400> 62
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 63
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V8
<400> 63
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 64
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V9
<400> 64
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 65
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V9
<400> 65
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 66
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V9
<400> 66
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 67
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V9
<400> 67
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 68
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V10
<400> 68
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Asn Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 69
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V10
<400> 69
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Asn Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 70
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V10
<400> 70
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 71
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V10
<400> 71
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 72
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V11
<400> 72
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 73
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V11
<400> 73
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 74
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V11
<400> 74
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Thr Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 75
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V11
<400> 75
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Thr Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 76
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V12
<400> 76
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Asn Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 77
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V12
<400> 77
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Asn Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 78
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V12
<400> 78
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Thr Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 79
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V12
<400> 79
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Arg Gly Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Thr Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 80
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VH_V13
<400> 80
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 81
<211> 445
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_HC_V13
<400> 81
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Asp Thr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val Asp His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys
210 215 220
Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly Pro Ser Val Phe Leu
225 230 235 240
Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu
245 250 255
Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln
260 265 270
Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys
275 280 285
Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu
290 295 300
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
305 310 315 320
Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys
325 330 335
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
340 345 350
Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
355 360 365
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
370 375 380
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
385 390 395 400
Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln
405 410 415
Glu Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
420 425 430
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Lys
435 440 445
<210> 82
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_VL_V13
<400> 82
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 83
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> HzVSF_LC_V13
<400> 83
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Val Ala Asp Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 84
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF hVL.hCkappa
<400> 84
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcaacaaact tagcagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgcacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 85
<211> 1335
<212> DNA
<213> Artificial Sequence
<220>
<223> hVH.hCgamma2
<400> 85
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tactacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcctcc 360
accaagggcc catcggtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gcggccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgctc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcaact tcggcaccca gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agacagttga gcgcaaatgt 660
tgtgtcgagt gcccaccgtg cccagcacca cctgtggcag gaccgtcagt cttcctcttc 720
cccccaaaac ccaaggacac cctcatgatc tcccggaccc ctgaggtcac gtgcgtggtg 780
gtggacgtga gccacgaaga ccccgaggtc cagttcaact ggtacgtgga cggcgtggag 840
gtgcataatg ccaagacaaa gccacgggag gagcagttca acagcacgtt ccgtgtggtc 900
agcgtcctca ccgtcgtgca ccaggactgg ctgaacggca aggagtacaa gtgcaaggtc 960
tccaacaaag gcctcccagc ccccatcgag aaaaccatct ccaaaaccaa agggcagccc 1020
cgagaaccac aggtgtacac cctgccccca tcccgggagg agatgaccaa gaaccaggtc 1080
agcctgacct gcctggtcaa aggcttctac cccagcgaca tcgccgtgga gtgggagagc 1140
aatgggcagc cggagaacaa ctacaacacc acacctccca tgctggactc cgacggctcc 1200
ttcttcctct acagcaagct caccgtggac aagagcaggt ggcagcaggg gaacgtcttc 1260
tcatgctccg tgatgcatga ggctctgcac aaccactaca cgcagaagag cctctccctg 1320
tctccgggta aatga 1335
<210> 86
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> hVH.hCgamma4
<400> 86
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tactacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 87
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF a1 gamma4
<400> 87
gagatccagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tactacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcctcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 88
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF a2 gamma4
<400> 88
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gattggaaat attgatcctt actatggtag tactacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcctcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 89
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF a3 gamma4
<400> 89
gagatccagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gattggaaat attgatcctt actatggtag tactacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcctcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 90
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> LC1
<400> 90
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcagacaact tagcagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 91
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> LC4
<400> 91
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcagacaacc gcggagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggta cccctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 92
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<223> VH1
<400> 92
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tgatacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctca 354
<210> 93
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<223> VH4
<400> 93
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tgatacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca cttccgcaag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggaataggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctca 354
<210> 94
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V1_HC
<400> 94
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tactacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 95
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V1_LC
<400> 95
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcagacaact tagcagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 96
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V2_HC
<400> 96
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tactacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 97
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V2_LC
<400> 97
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcagacaact taggagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 98
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V3_HC
<400> 98
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tactacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 99
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V3_LC
<400> 99
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcagacaacc gcggagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 100
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V4_HC
<400> 100
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tactacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 101
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V4_LC
<400> 101
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcagacaacc gcggagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggta cccctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 102
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V5_HC
<400> 102
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tgatacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 103
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V5_LC
<400> 103
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcaacaaact tagcagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 104
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V6_HC
<400> 104
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tactacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca cttccgcaag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 105
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V6_LC
<400> 105
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcaacaaact tagcagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 106
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V7_HC
<400> 106
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tgatacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca cwtccgsmag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 107
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V7_LC
<400> 107
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcaacaaact tagcagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 108
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V8_HC
<400> 108
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tgatacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca cttccgcaag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggaataggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 109
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V8_LC
<400> 109
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcaacaaact tagcagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 110
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V9_HC
<400> 110
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tgatacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca cttccgcaag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 111
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V9_LC
<400> 111
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcagacaacc gcggagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 112
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V10_HC
<400> 112
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tgatacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca cttccgcaag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggaataggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 113
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V10_LC
<400> 113
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcagacaacc gcggagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 114
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V11_HC
<400> 114
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tgatacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca cttccgcaag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 115
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V11_LC
<400> 115
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcagacaacc gcggagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggta cccctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 116
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V12_HC
<400> 116
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tgatacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca cttccgcaag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggaataggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 117
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V12_LC
<400> 117
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcagacaacc gcggagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggta cccctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 118
<211> 1338
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V13_HC
<400> 118
gaggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctacaaca tgaactgggt gcgacaggct 120
cctggaaaag ggcttgagtg gatgggaaat attgatcctt actatggtag tgatacctat 180
gcacagaagt ttcagggcag ggtcaccatg accgtagaca aatccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaaccctgg tcaccgtctc ctcagcttcc 360
accaagggcc catccgtctt ccccctggcg ccctgctcca ggagcacctc cgagagcaca 420
gccgccctgg gctgcctggt caaggactac ttccccgaac cggtgacggt gtcgtggaac 480
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 540
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcacgaa gacctacacc 600
tgcaacgtag atcacaagcc cagcaacacc aaggtggaca agagagttga gtccaaatat 660
ggtcccccat gcccatcatg cccagcacct gagttcctgg ggggaccatc agtcttcctg 720
ttccccccaa aacccaagga cactctcatg atctcccgga cccctgaggt cacgtgcgtg 780
gtggtggacg tgagccagga agaccccgag gtccagttca actggtacgt ggatggcgtg 840
gaggtgcata atgccaagac aaagccgcgg gaggagcagt tcaacagcac gtaccgtgtg 900
gtcagcgtcc tcaccgtcct gcaccaggac tggctgaacg gcaaggagta caagtgcaag 960
gtctccaaca aaggcctccc gtcctccatc gagaaaacca tctccaaagc caaagggcag 1020
ccccgagagc cacaggtgta caccctgccc ccatcccagg aggagatgac caagaaccag 1080
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1140
agcaatgggc agccggagaa caactacaag accacgcctc ccgtgctgga ctccgacggc 1200
tccttcttcc tctacagcag gctaaccgtg gacaagagca ggtggcagga ggggaatgtc 1260
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacacagaa gagcctctcc 1320
ctgtctctgg gtaaatga 1338
<210> 119
<211> 645
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF_V13_LC
<400> 119
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacca 120
gggaaagctc ctaagctcct gatctatgtt gcagacaact tagcagatgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacat ttttatggtt ctcctcggac gttcggcgga 300
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 360
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 420
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 480
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 540
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 600
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 645
<210> 120
<211> 1417
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF V12_HC_оптимизация
<400> 120
aagcttgccg ccaccatgga atggtcctgg gtgttcctgt tcttcctgtc cgtgaccacc 60
ggcgtgcact ccgaagtgca gctggtgcag tctggcgccg aagtgaagaa acctggcgcc 120
tccgtgaagg tgtcctgcaa ggcttccggc tacaccttta ccggctacaa catgaactgg 180
gtgcgacagg cccctggcaa gggcctggaa tggatgggca acatcgaccc ctactacggc 240
tccgacacct acgcccagaa attccagggc agagtgacca tgaccgtgga cacctctgcc 300
tccaccgcct acatggaact gtcccggctg agatccgacg acaccgccgt gtactactgc 360
gccagagaga caggcaaccg ggccatggat tattggggcc agggcaccct cgtgaccgtg 420
tctagcgctt ctaccaaggg cccctccgtg ttccctctgg ccccttgctc cagatccacc 480
tccgagtcta ccgccgctct gggctgcctc gtgaaggact acttccccga gcccgtgaca 540
gtgtcctgga actctggcgc tctgacctct ggcgtgcaca ccttccctgc tgtgctgcag 600
tcctccggcc tgtactccct gtcctccgtc gtgactgtgc cctccagctc tctgggcacc 660
aagacctaca cctgtaacgt ggaccacaag ccctccaaca ccaaggtgga caagcgggtg 720
gaatctaagt acggccctcc ctgccctagc tgccctgccc ctgagtttct gggaggccct 780
tctgtgtttc tgttcccccc aaagcccaag gacaccctga tgatctcccg gacccccgaa 840
gtgacctgcg tggtggtgga tgtgtcccag gaagatcccg aggtgcagtt caattggtac 900
gtggacggcg tggaagtgca caacgccaag accaagccta gagaggaaca gttcaactcc 960
acctaccggg tggtgtccgt gctgaccgtg ctgcaccagg attggctgaa cggcaaagag 1020
tacaagtgca aggtgtccaa caagggactg cccagctcca tcgaaaagac catctccaag 1080
gccaagggcc agccccggga accccaggtg tacacactgc ctccaagcca ggaagagatg 1140
accaagaacc aggtgtccct gacctgtctc gtgaaaggct tctacccctc cgatatcgcc 1200
gtggaatggg agtccaacgg ccagcctgag aacaactaca agaccacccc ccctgtgctg 1260
gactccgacg gctccttctt tctgtactct cggctgacag tggacaagtc ccggtggcag 1320
gaaggcaacg tgttctcctg cagcgtgatg cacgaggccc tgcacaacca ctacacccag 1380
aagtccctgt ccctgtctct gggaaagtga tgaattc 1417
<210> 121
<211> 727
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF V12_LC_ оптимизация
<400> 121
aagcttgccg ccaccatgtc cgtgcctacc caggtgctgg gactgctgct gctgtggctg 60
accgacgcca gatgcgacat ccagatgacc cagtccccct ccagcctgtc tgcttccgtg 120
ggcgacagag tgaccatcac ctgtcgggcc tccgagaaca tctactccaa cctggcctgg 180
tatcagcaga agcccggcaa ggcccccaag ctgctgatct acgtggccga caatagaggc 240
gacggcgtgc cctccagatt ctccggctct ggctctggca ccgactttac cctgaccatc 300
agctccctgc agcccgagga cttcgccacc tactactgcc agcacttcta cggcaccccc 360
cggacatttg gcggaggcac caaggtggaa atcaagcgga ccgtggccgc tccctccgtg 420
ttcatcttcc caccttccga cgagcagctg aagtccggca ccgcttctgt cgtgtgcctg 480
ctgaacaact tctacccccg cgaggccaag gtgcagtgga aggtggacaa cgccctgcag 540
tccggcaact cccaggaatc cgtgaccgag caggactcca aggacagcac ctactccctg 600
tcctccaccc tgaccctgtc caaggccgac tacgagaagc acaaggtgta cgcctgcgaa 660
gtgacccacc agggcctgtc tagccccgtg accaagtctt tcaaccgggg cgagtgctga 720
tgaattc 727
<210> 122
<211> 1417
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF V13_HC_оптимизация
<400> 122
aagcttgccg ccaccatgga atggtcctgg gtgttcctgt tcttcctgtc cgtgaccacc 60
ggcgtgcact ccgaagtgca gctggtgcag tctggcgccg aagtgaagaa acctggcgcc 120
tccgtgaagg tgtcctgcaa ggcttccggc tacaccttta ccggctacaa catgaactgg 180
gtgcgacagg cccctggcaa gggcctggaa tggatgggca acatcgaccc ctactacggc 240
tccgacacct acgcccagaa attccagggc agagtgacca tgaccgtgga caagtccatc 300
tccaccgcct acatggaact gtcccggctg agatccgacg acaccgccgt gtactactgc 360
gccagagaga caggcacccg ggccatggat tattggggcc agggcaccct cgtgaccgtg 420
tcctctgctt ctaccaaggg cccctccgtg ttccctctgg ccccttgctc cagatccacc 480
tccgagtcta ccgccgctct gggctgcctc gtgaaggact acttccccga gcccgtgaca 540
gtgtcttgga actctggcgc cctgacctct ggcgtgcaca cctttccagc tgtgctgcag 600
tcctccggcc tgtactccct gtcctccgtc gtgactgtgc cctccagctc tctgggcacc 660
aagacctaca cctgtaacgt ggaccacaag ccctccaaca ccaaggtgga caagcgggtg 720
gaatctaagt acggccctcc ctgccctagc tgccctgccc ctgagtttct gggaggccct 780
tctgtgtttc tgttcccccc aaagcccaag gacaccctga tgatctcccg gacccccgaa 840
gtgacctgcg tggtggtgga tgtgtcccag gaagatcccg aggtgcagtt caattggtac 900
gtggacggcg tggaagtgca caacgccaag accaagccta gagaggaaca gttcaactcc 960
acctaccggg tggtgtccgt gctgaccgtg ctgcaccagg attggctgaa cggcaaagag 1020
tacaagtgca aggtgtccaa caagggactg cccagctcca tcgaaaagac catctccaag 1080
gccaagggcc agccccggga accccaggtg tacacactgc ctccaagcca ggaagagatg 1140
accaagaacc aggtgtccct gacctgtctc gtgaaaggct tctacccctc cgatatcgcc 1200
gtggaatggg agtccaacgg ccagcctgag aacaactaca agaccacccc ccctgtgctg 1260
gactccgacg gctccttctt tctgtactct cggctgacag tggataagtc ccggtggcag 1320
gaaggcaacg tgttctcctg cagcgtgatg cacgaggccc tgcacaacca ctacacccag 1380
aagtccctgt ccctgtctct gggaaagtga tgaattc 1417
<210> 123
<211> 727
<212> DNA
<213> Artificial Sequence
<220>
<223> HzVSF V13_LC_оптимизация
<400> 123
aagcttgccg ccaccatgtc cgtgcctacc caggtgctgg gactgctgct gctgtggctg 60
accgacgcca gatgcgacat ccagatgacc cagtccccct ccagcctgtc tgcttccgtg 120
ggcgacagag tgaccatcac ctgtcgggcc tccgagaaca tctactccaa cctggcctgg 180
tatcagcaga agcccggcaa ggcccccaag ctgctgatct acgtggccga taacctggcc 240
gacggcgtgc cctctagatt ctccggctct ggctctggca ccgactttac cctgaccatc 300
agctccctgc agcccgagga cttcgccacc tactactgcc agcacttcta cggctccccc 360
cggacatttg gcggaggcac caaggtggaa atcaagcgga ccgtggccgc tccctccgtg 420
ttcatcttcc caccttccga cgagcagctg aagtccggca ccgcttctgt cgtgtgcctg 480
ctgaacaact tctacccccg cgaggccaag gtgcagtgga aggtggacaa cgccctgcag 540
tccggcaact cccaggaatc cgtgaccgag caggactcca aggacagcac ctactccctg 600
tcctccaccc tgaccctgtc caaggccgac tacgagaagc acaaggtgta cgcctgcgaa 660
gtgacccacc agggcctgtc tagccccgtg accaagtctt tcaaccgggg cgagtgctga 720
tgaattc 727
<210> 124
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер mVH F
<400> 124
cgagctcatg ggatggagct ggatc 25
<210> 125
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер mVH R
<400> 125
cggtacctga ggagacggtg actg 24
<210> 126
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> KpnI_delR праймер
<400> 126
gggcccttgg tggaagctga ggagacggtg actgagg 37
<210> 127
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер mVL F
<400> 127
catcgatatg agtgtgccca ctcag 25
<210> 128
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер mVL R
<400> 128
cctcgagttt gatttccagc ttgg 24
<210> 129
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> Xho_modR праймер
<400> 129
agatggtgca gccaccgtgc gtttgatttc cagcttggtg cc 42
<210> 130
<211> 354
<212> DNA
<213> Artificial Sequence
<220>
<223> ScFv VSF H
<400> 130
gagatccagc tgcagcagtc tggagctgag ctggtgaagc ctggggcttc agtgaagata 60
tcctgcaagg cttctggtta ctcattcact ggctacaaca tgaactgggt gaagcagagc 120
catggaaaga gccttgagtg gattggaaat attgatcctt actatggtag tactacctac 180
aatcagaagt tcaagggcaa ggccacattg actgtagaca aatcttccag cacagcctac 240
atgcagctca acagcctgac atctgaggac tctgcagtct attactgtgc aagagagact 300
gggacgaggg ctatggacta ctggggtcaa ggaacctcag tcaccgtctc ctca 354
<210> 131
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> scFv VSF H
<400> 131
Glu Ile Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ser Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Lys Gln Ser His Gly Lys Ser Leu Glu Trp Ile
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Asn Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Ser Val Thr Val Ser Ser
115
<210> 132
<211> 321
<212> DNA
<213> Artificial Sequence
<220>
<223> scFv VSF L3
<400> 132
gacatccaga tgactcagtc tccagcctcc ctatctgcat ctgtgggaga aactgtcacc 60
atgacatgtc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacag 120
ggaaaatctc ctcagctcct ggtctatgtt gcaacaaact tagcagatgg tgtgccatca 180
aggttcagtg gcagtggatc aggcacacag ttttctctga agatcaacag cctgcagcct 240
gaagattttg ggagttatta ctgtcaacat ttttatggtt ctcctcggac gttcggtgga 300
ggcaccaagc tggaaatcaa a 321
<210> 133
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> scFv VSF L3
<400> 133
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Glu Thr Val Thr Met Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro Gln Leu Leu Val
35 40 45
Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Phe Ser Leu Lys Ile Asn Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Ser Tyr Tyr Cys Gln His Phe Tyr Gly Ser Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 134
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> mVSF CDR-L1
<400> 134
Arg Ala Ser Glu Asn Ile Tyr Ser Asn Leu Ala Trp Tyr
1 5 10
<210> 135
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> mVSF CDR-L2
<400> 135
Leu Leu Val Tyr Val Ala Thr Asn Leu Ala Asp
1 5 10
<210> 136
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> mVSF CDR-L3
<400> 136
Gln His Phe Tyr Gly Ser Pro Arg Thr
1 5
<210> 137
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> mVSF CDR-H1
<400> 137
Gly Tyr Ser Phe Thr Gly Tyr Asn Met Asn
1 5 10
<210> 138
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> mVSF CDR-H2
<400> 138
Trp Ile Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Asn Gln
1 5 10 15
Lys Phe Lys Gly
20
<210> 139
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> mVSF CDR-H3
<400> 139
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr
1 5 10
<210> 140
<211> 154
<212> PRT
<213> Artificial Sequence
<220>
<223> chVSFk
<400> 140
Met Ser Val Pro Thr Gln Val Leu Gly Leu Leu Leu Leu Trp Leu Thr
1 5 10 15
Gly Ala Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser
20 25 30
Ala Ser Val Gly Glu Thr Val Thr Met Thr Cys Arg Ala Ser Glu Asn
35 40 45
Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro
50 55 60
Gln Leu Leu Val Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser
65 70 75 80
Arg Phe Ser Gly Ser Gly Ser Gly Thr Gln Phe Ser Leu Lys Ile Asn
85 90 95
Ser Leu Gln Pro Glu Asp Phe Gly Ser Tyr Tyr Cys Gln His Phe Tyr
100 105 110
Gly Ser Pro Arg Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
115 120 125
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
130 135 140
Leu Lys Ser Gly Thr Ala Ser Val Val Cys
145 150
<210> 141
<211> 142
<212> PRT
<213> Artificial Sequence
<220>
<223> chVSFg4
<400> 141
Met Gly Trp Ser Trp Ile Phe Leu Phe Leu Leu Ser Val Thr Ala Gly
1 5 10 15
Val His Ser Glu Ile Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Lys
20 25 30
Pro Gly Ala Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ser Phe
35 40 45
Thr Gly Tyr Asn Met Asn Trp Val Lys Gln Ser His Gly Lys Ser Leu
50 55 60
Glu Trp Ile Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Asn
65 70 75 80
Gln Lys Phe Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser
85 90 95
Thr Ala Tyr Met Gln Leu Asn Ser Leu Thr Ser Glu Asp Ser Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Asn Gly
115 120 125
Gln Gly Thr Ser Val Thr Val Ser Ser Gly Thr Ala Ser Thr
130 135 140
<210> 142
<211> 131
<212> PRT
<213> Artificial Sequence
<220>
<223> chVSFg2
<400> 142
Glu Ile Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ser Phe Thr Gly Tyr
20 25 30
Asn Met Asn Trp Val Lys Gln Ser His Gly Lys Ser Leu Glu Trp Ile
35 40 45
Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Asn Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Ser Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro
130
<210> 143
<211> 234
<212> PRT
<213> Artificial Sequence
<220>
<223> Легкая цепь chVSF
<400> 143
Met Ser Val Pro Thr Gln Val Leu Gly Leu Leu Leu Leu Trp Leu Thr
1 5 10 15
Gly Ala Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser
20 25 30
Ala Ser Val Gly Glu Thr Val Thr Met Thr Cys Arg Ala Ser Glu Asn
35 40 45
Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro
50 55 60
Gln Leu Leu Val Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser
65 70 75 80
Arg Phe Ser Gly Ser Gly Ser Gly Thr Gln Phe Ser Leu Lys Ile Asn
85 90 95
Ser Leu Gln Pro Glu Asp Phe Gly Ser Tyr Tyr Cys Gln His Phe Tyr
100 105 110
Gly Ser Pro Arg Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
115 120 125
Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
130 135 140
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
145 150 155 160
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
165 170 175
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
180 185 190
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
195 200 205
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
210 215 220
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230
<210> 144
<211> 234
<212> PRT
<213> Artificial Sequence
<220>
<223> hCk - mVL
<400> 144
Met Ser Val Pro Thr Gln Val Arg Gly Leu Leu Leu Leu Trp Leu Thr
1 5 10 15
Gly Ala Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser
20 25 30
Ala Ser Val Gly Glu Thr Val Thr Met Thr Cys Arg Ala Ser Glu Asn
35 40 45
Ile Tyr Ser Asn Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro
50 55 60
Gln Leu Leu Val Tyr Val Ala Thr Asn Leu Ala Asp Gly Val Pro Ser
65 70 75 80
Arg Phe Ser Gly Ser Gly Ser Gly Thr Gln Phe Ser Leu Met Ile Asn
85 90 95
Ser Leu Gln Pro Glu Asp Phe Gly Ser Tyr Tyr Cys Gln His Phe Tyr
100 105 110
Gly Pro Pro Arg Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Leu
115 120 125
Glu Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln
130 135 140
Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr
145 150 155 160
Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser
165 170 175
Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr
180 185 190
Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys
195 200 205
His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro
210 215 220
Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230
<210> 145
<211> 705
<212> DNA
<213> Artificial Sequence
<220>
<223> hCk - mVL
<400> 145
atgagtgtgc ccactcaggt ccgggggttg ctgctgctgt ggcttacagg tgccagatgt 60
gacatccaga tgactcagtc tccagcctcc ctatctgcat ctgtgggaga aactgtcacc 120
atgacatgtc gagcaagtga gaatatttac agtaatttag catggtatca gcagaaacag 180
ggaaaatctc ctcagctcct ggtctatgtt gcaacaaact tagcagatgg tgtgccatca 240
aggttcagtg gcagtggatc aggcacacag ttttctctga tgatcaacag cctgcagcct 300
gaagattttg ggagttatta ctgtcaacat ttttatggtc ctcctcggac gttcggtgga 360
ggcaccaagc tggaaatcaa actcgaggtg gctgcaccat ctgtcttcat cttcccgcca 420
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 480
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 540
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 600
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 660
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gttag 705
<210> 146
<211> 465
<212> PRT
<213> Artificial Sequence
<220>
<223> hCr2 - mVH
<400> 146
Met Gly Trp Ser Trp Ile Phe Leu Phe Leu Leu Ser Val Thr Ala Gly
1 5 10 15
Val His Ser Glu Ile Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Lys
20 25 30
Pro Gly Ala Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ser Phe
35 40 45
Thr Gly Tyr Asn Met Asn Trp Val Lys Gln Ser His Gly Lys Ser Leu
50 55 60
Glu Trp Ile Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Asn
65 70 75 80
Gln Lys Phe Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser
85 90 95
Thr Ala Tyr Met Gln Leu Asn Ser Leu Thr Ser Glu Asp Ser Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly
115 120 125
Gln Gly Thr Ser Val Thr Val Ser Ser Gly Thr Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr Tyr Thr Cys Asn Val
210 215 220
Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Thr Val Glu Arg Lys
225 230 235 240
Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro Pro Val Ala Gly Pro
245 250 255
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
260 265 270
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
275 280 285
Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
290 295 300
Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Phe Arg Val
305 310 315 320
Val Ser Val Leu Thr Val Val His Gln Asp Trp Leu Asn Gly Lys Glu
325 330 335
Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ala Pro Ile Glu Lys
340 345 350
Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
355 360 365
Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
370 375 380
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
385 390 395 400
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Asn Thr Thr Pro Pro Met Leu
405 410 415
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
420 425 430
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
435 440 445
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
450 455 460
Lys
465
<210> 147
<211> 1398
<212> DNA
<213> Artificial Sequence
<220>
<223> hCr2 - mVH
<400> 147
atgggatgga gctggatctt tctcttcctt ctgtcagtaa ctgcaggtgt ccactctgag 60
atccagctgc agcagtctgg agctgagctg gtgaagcctg gggcttcagt gaagatatcc 120
tgcaaggctt ctggttactc attcactggc tacaacatga actgggtgaa gcagagccat 180
ggaaagagcc ttgagtggat tggaaatatt gatccttact atggtagtac tacctacaat 240
cagaagttca agggcaaggc cacattgact gtagacaaat cttccagcac agcctacatg 300
cagctcaaca gcctgacatc tgaggactct gcagtctatt actgtgcaag agagactggg 360
acgagggcta tggactactg gggtcaagga acctcagtca ccgtctcctc aggtaccgcc 420
tccaccaagg gcccatcggt cttccccctg gcgccctgct ccaggagcac ctccgagagc 480
acagcggccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 540
aactcaggcg ctctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 600
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca acttcggcac ccagacctac 660
acctgcaacg tagatcacaa gcccagcaac accaaggtgg acaagacagt tgagcgcaaa 720
tgttgtgtcg agtgcccacc gtgcccagca ccacctgtgg caggaccgtc agtcttcctc 780
ttccccccaa aacccaagga caccctcatg atctcccgga cccctgaggt cacgtgcgtg 840
gtggtggacg tgagccacga agaccccgag gtccagttca actggtacgt ggacggcgtg 900
gaggtgcata atgccaagac aaagccacgg gaggagcagt tcaacagcac gttccgtgtg 960
gtcagcgtcc tcaccgtcgt gcaccaggac tggctgaacg gcaaggagta caagtgcaag 1020
gtctccaaca aaggcctccc agcccccatc gagaaaacca tctccaaaac caaagggcag 1080
ccccgagaac cacaggtgta caccctgccc ccatcccggg aggagatgac caagaaccag 1140
gtcagcctga cctgcctggt caaaggcttc taccccagcg acatcgccgt ggagtgggag 1200
agcaatgggc agccggagaa caactacaac accacacctc ccatgctgga ctccgacggc 1260
tccttcttcc tctacagcaa gctcaccgtg gacaagagca ggtggcagca ggggaacgtc 1320
ttctcatgct ccgtgatgca tgaggctctg cacaaccact acacgcagaa gagcctctcc 1380
ctgtctccgg gtaaatga 1398
<210> 148
<211> 466
<212> PRT
<213> Artificial Sequence
<220>
<223> hCr4 - mVH
<400> 148
Met Gly Trp Ser Trp Ile Phe Leu Phe Leu Leu Ser Val Thr Ala Gly
1 5 10 15
Val His Ser Glu Ile Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Lys
20 25 30
Pro Gly Ala Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Ser Phe
35 40 45
Thr Gly Tyr Asn Met Asn Trp Val Lys Gln Ser His Gly Lys Ser Leu
50 55 60
Glu Trp Ile Gly Asn Ile Asp Pro Tyr Tyr Gly Ser Thr Thr Tyr Asn
65 70 75 80
Gln Lys Phe Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser
85 90 95
Thr Ala Tyr Met Gln Leu Asn Ser Leu Thr Ser Glu Asp Ser Ala Val
100 105 110
Tyr Tyr Cys Ala Arg Glu Thr Gly Thr Arg Ala Met Asp Tyr Trp Gly
115 120 125
Gln Gly Thr Ser Val Thr Val Ser Ser Gly Thr Ala Ser Thr Lys Gly
130 135 140
Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg Ser Thr Ser Glu Ser
145 150 155 160
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
165 170 175
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
180 185 190
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
195 200 205
Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr Tyr Thr Cys Asn Val
210 215 220
Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Ser Lys
225 230 235 240
Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe Leu Gly Gly
245 250 255
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
260 265 270
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu
275 280 285
Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
290 295 300
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg
305 310 315 320
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
325 330 335
Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu
340 345 350
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
355 360 365
Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu
370 375 380
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
385 390 395 400
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
405 410 415
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp
420 425 430
Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Met His
435 440 445
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu
450 455 460
Gly Lys
465
<210> 149
<211> 1401
<212> DNA
<213> Artificial Sequence
<220>
<223> hCr4 - mVH
<400> 149
atgggatgga gctggatctt tctcttcctt ctgtcagtaa ctgcaggtgt ccactctgag 60
atccagctgc agcagtctgg agctgagctg gtgaagcctg gggcttcagt gaagatatcc 120
tgcaaggctt ctggttactc attcactggc tacaacatga actgggtgaa gcagagccat 180
ggaaagagcc ttgagtggat tggaaatatt gatccttact atggtagtac tacctacaat 240
cagaagttca agggcaaggc cacattgact gtagacaaat cttccagcac agcctacatg 300
cagctcaaca gcctgacatc tgaggactct gcagtctatt actgtgcaag agagactggg 360
acgagggcta tggactactg gggtcaagga acctcagtca ccgtctcctc aggtaccgct 420
tccaccaagg gcccatccgt cttccccctg gcgccctgct ccaggagcac ctccgagagc 480
acagccgccc tgggctgcct ggtcaaggac tacttccccg aaccggtgac ggtgtcgtgg 540
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 600
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac gaagacctac 660
acctgcaacg tagatcacaa gcccagcaac accaaggtgg acaagagagt tgagtccaaa 720
tatggtcccc catgcccatc atgcccagca cctgagttcc tggggggacc atcagtcttc 780
ctgttccccc caaaacccaa ggacactctc atgatctccc ggacccctga ggtcacgtgc 840
gtggtggtgg acgtgagcca ggaagacccc gaggtccagt tcaactggta cgtggatggc 900
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agttcaacag cacgtaccgt 960
gtggtcagcg tcctcaccgt cctgcaccag gactggctga acggcaagga gtacaagtgc 1020
aaggtctcca acaaaggcct cccgtcctcc atcgagaaaa ccatctccaa agccaaaggg 1080
cagccccgag agccacaggt gtacaccctg cccccatccc aggaggagat gaccaagaac 1140
caggtcagcc tgacctgcct ggtcaaaggc ttctacccca gcgacatcgc cgtggagtgg 1200
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1260
ggctccttct tcctctacag caggctaacc gtggacaaga gcaggtggca ggaggggaat 1320
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacaca gaagagcctc 1380
tccctgtctc tgggtaaatg a 1401
<210> 150
<211> 851
<212> DNA
<213> Artificial Sequence
<220>
<223> ДНК scFv VSF
<400> 150
cgtcgacgag atccagctgc agcagtctga gatccagctg cagcagtctg gagctgagct 60
ggtgaagcct ggggcttcag tgaagatatc ctgcaaggct tctggttact cattcactgg 120
ctacaacatg aactgggtga agcagagcca tggaaagagc cttgagtgga ttggaaatat 180
tgatccttac tatggtagta ctacctacaa tcagaagttc aagggcaagg ccacattgac 240
tgtagacaaa tcttccagca cagcctacat gcagctcaac agcctgacat ctgaggactc 300
tgcagtctat tactgtgcaa gagagactgg gacgagggct atggactact ggggtcaagg 360
aacctcagtc accgtctcct caccttggag tcagtggcag aggagtccac ctcacgagac 420
cacccctcct aggccacctc aggaggatcc ggtggaggtg gctctggtgg aggtggctct 480
gacatccaga tgactcagtc gacatccaga tgactcagtc tccagcctcc ctatctgcat 540
ctgtgggaga aactgtcacc atgacatgtc gagcaagtga gaatatttac agtaatttag 600
catggtatca gcagaaacag ggaaaatctc ctcagctcct ggtctatgtt gcaacaaact 660
tagcagatgg tgtgccatca aggttcagtg gcagtggatc aggcacacag ttttctctga 720
agatcaacag cctgcagcct gaagattttg ggagttatta ctgtcaacat ttttatggtt 780
ctcctcggac gttcggtgga ggcaccaagc tggaaatcaa acccgtggtt cgacctttag 840
tttggagctc c 851
<210> 151
<211> 30
<212> PRT
<213> Artificial Sequence
<220>
<223> FR1 тяжелой цепи
<400> 151
Glu Ile Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr
20 25 30
<210> 152
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> FR2 тяжелой цепи
<400> 152
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile Gly
1 5 10
<210> 153
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> HBV cccDNA праймер
<400> 153
tgaatccygc ggacgacc 18
<210> 154
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> HBV cccDNA праймер R
<400> 154
cagcttggag gcttgaacag 20
<210> 155
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер HBV total DNA F
<400> 155
cctcttcatc ctgctgct 18
<210> 156
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер HBV total DNA R
<400> 156
aactgaaagc caaacagtg 19
<210> 157
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер HCV target F
<400> 157
gggctataag gtgctagtgc 20
<210> 158
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер HCV target R
<400> 158
ggctgccagt ggtaattgtt 20
<210> 159
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер GAPDH F
<400> 159
tccctgagct gaacgggaag 20
<210> 160
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер GAPDH R
<400> 160
ggaggagtgg gtgtcgctgt 20
<210> 161
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер HCV target F-2
<400> 161
atgcagccca agggtataag 20
<210> 162
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Праймер HCV target R-2
<400> 162
ggttctgatg ttagggtcga tac 23
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Новый вариант О-сукцинилгомосеринтрансферазы и способ получения О-сукцинилгомосерина с использованием этого варианта | 2018 |
|
RU2747493C1 |
| АНТИТЕЛА ПРОТИВ БЕЛКА-1 ЗАПРОГРАММИРОВАННОЙ КЛЕТОЧНОЙ СМЕРТИ (PD-1) И ИХ ПРИМЕНЕНИЕ | 2017 |
|
RU2725950C1 |
| Новый мутант О-сукцинилгомосеринтрансферазы и способ получения О-сукцинилгомосерина с использованием этого мутанта | 2018 |
|
RU2747494C1 |
| НОВЫЕ ВАРИАНТЫ ГИАЛУРОНИДАЗЫ С УЛУЧШЕННОЙ СТАБИЛЬНОСТЬЮ И СОДЕРЖАЩАЯ ИХ ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ | 2021 |
|
RU2811464C2 |
| МУТИРОВАННЫЙ БЕЛОК SPOT И СПОСОБ ПОЛУЧЕНИЯ L-АМИНОКИСЛОТ С ЕГО ПРИМЕНЕНИЕМ | 2022 |
|
RU2837657C2 |
| Биспецифическое антитело анти-HER2/анти-4-1BB и его применение | 2020 |
|
RU2831838C2 |
| АНТИТЕЛА К C10ORF54 И ИХ ПРИМЕНЕНИЯ | 2015 |
|
RU2819627C1 |
| Новый вариант L-тирозин-экспортирующего белка и способ получения L-тирозина с его использованием | 2021 |
|
RU2808831C1 |
| БИСПЕЦИФИЧЕСКОЕ АНТИТЕЛО АНТИ-EGFR/АНТИ-4-1ВВ И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2831836C2 |
| Антитела против лиганда-1 запрограммированной смерти (PD-L1) и их применение | 2017 |
|
RU2721582C1 |
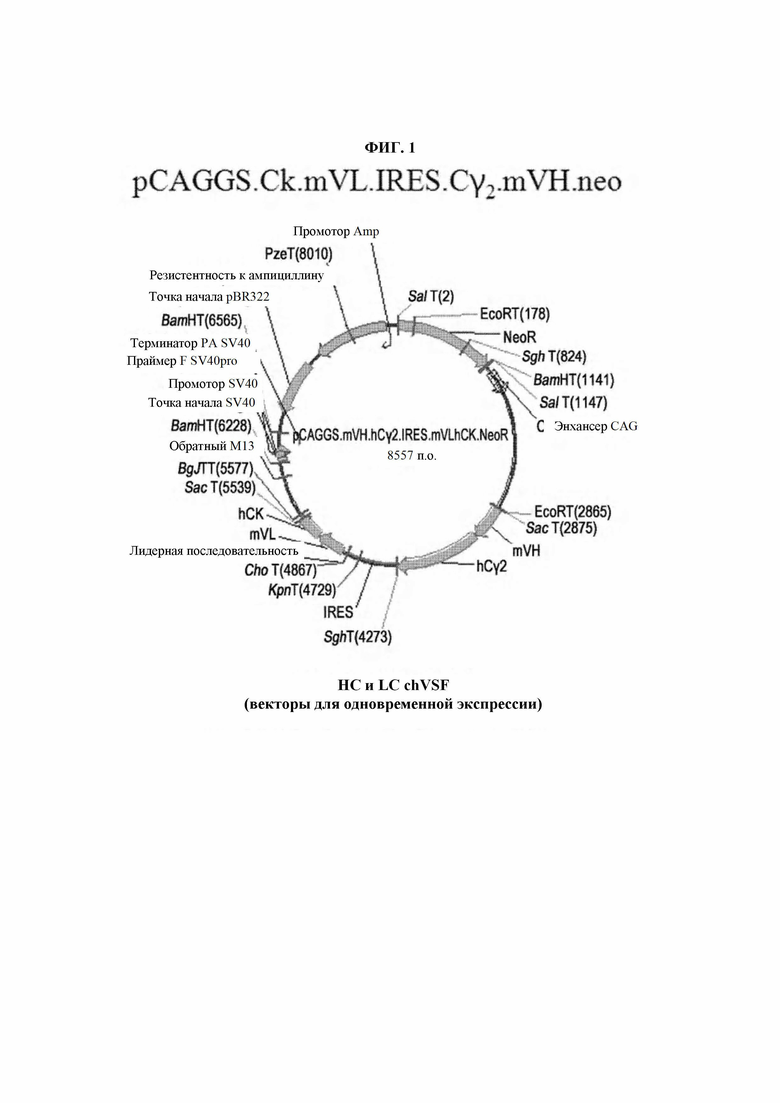
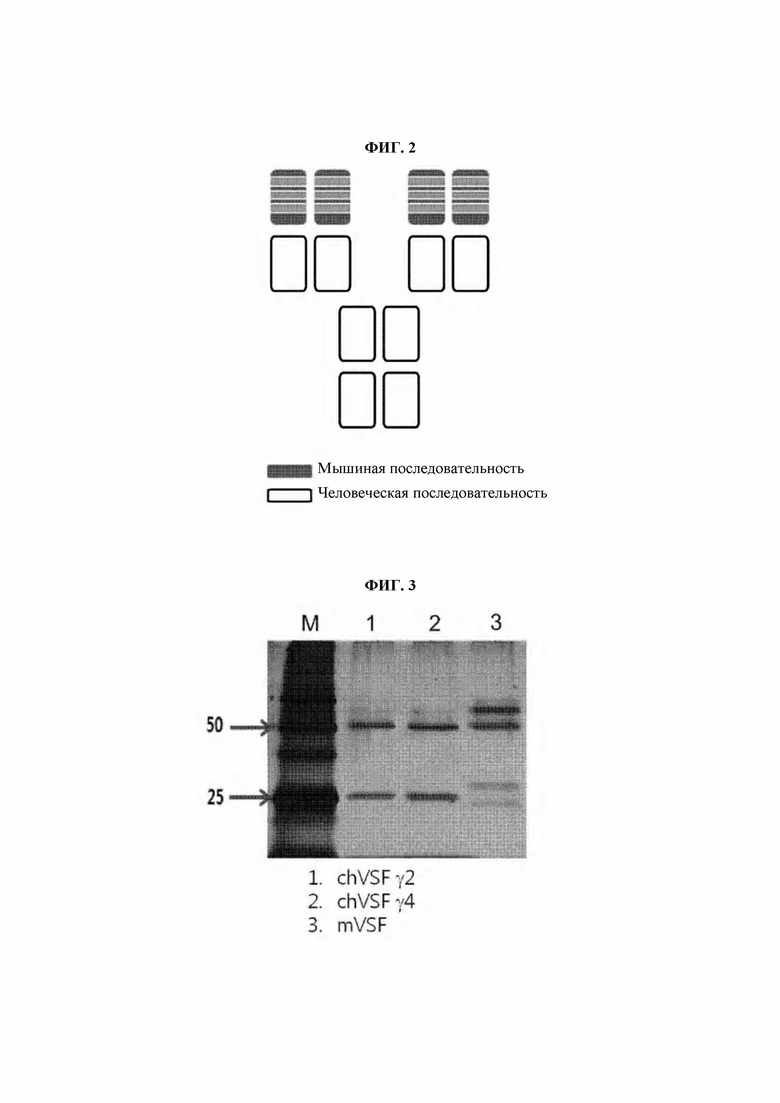

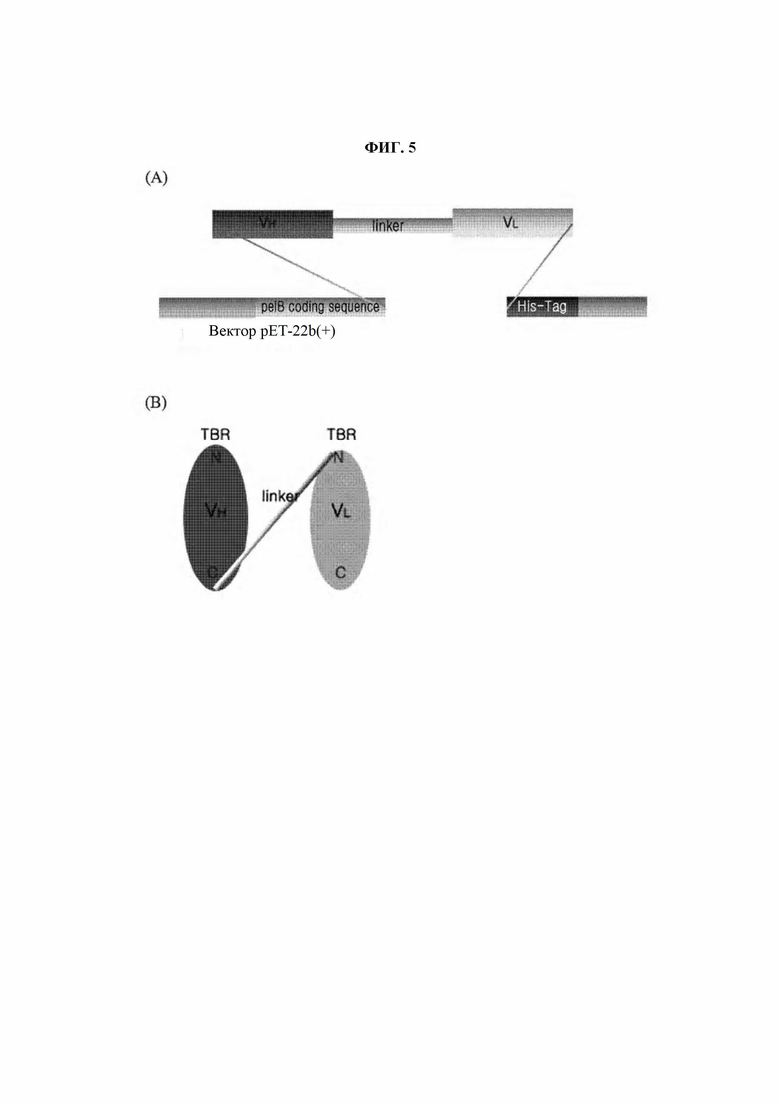
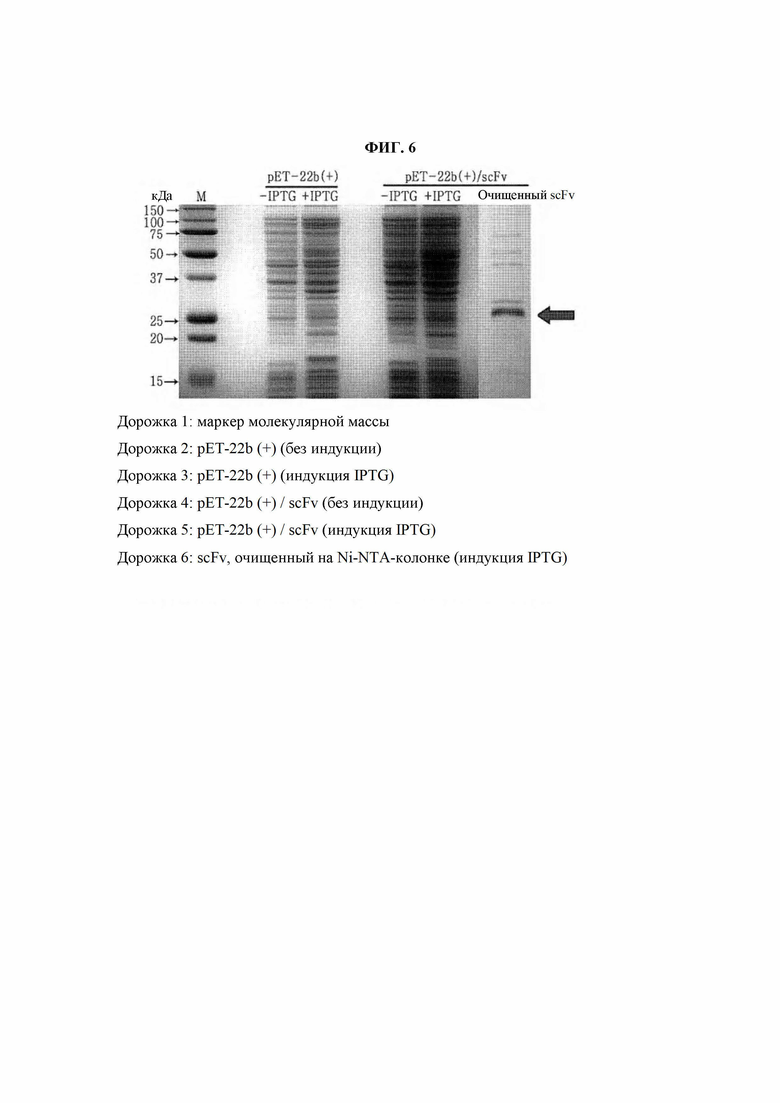
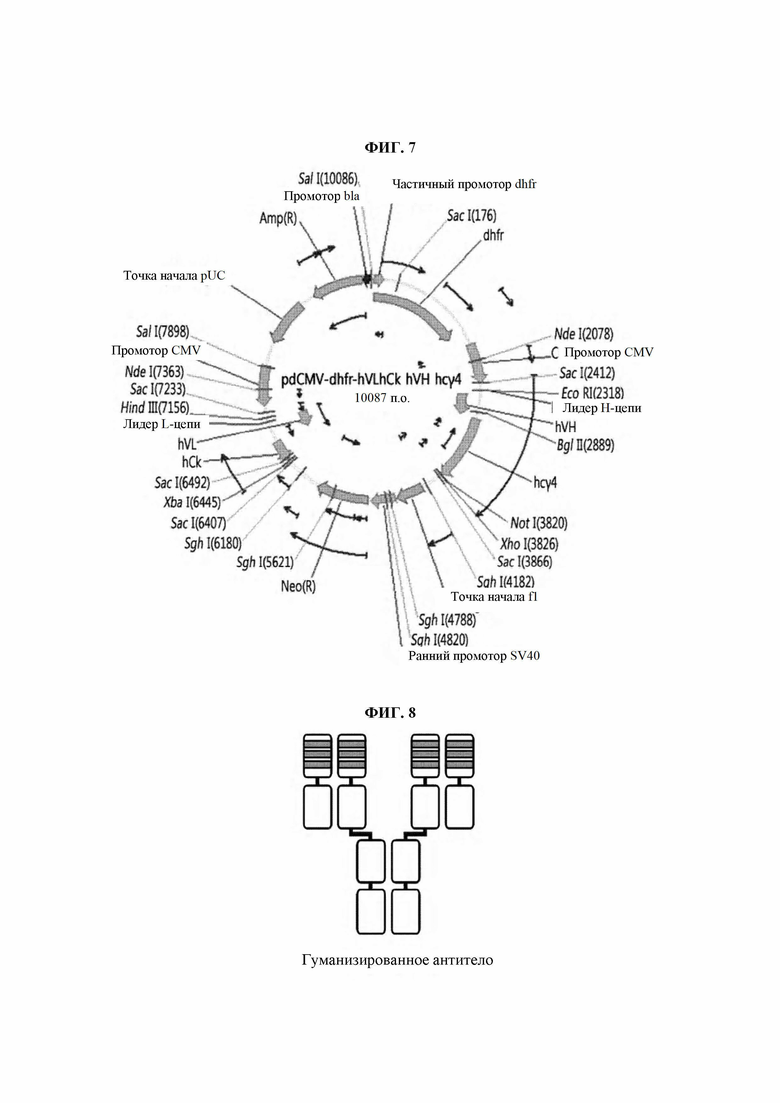
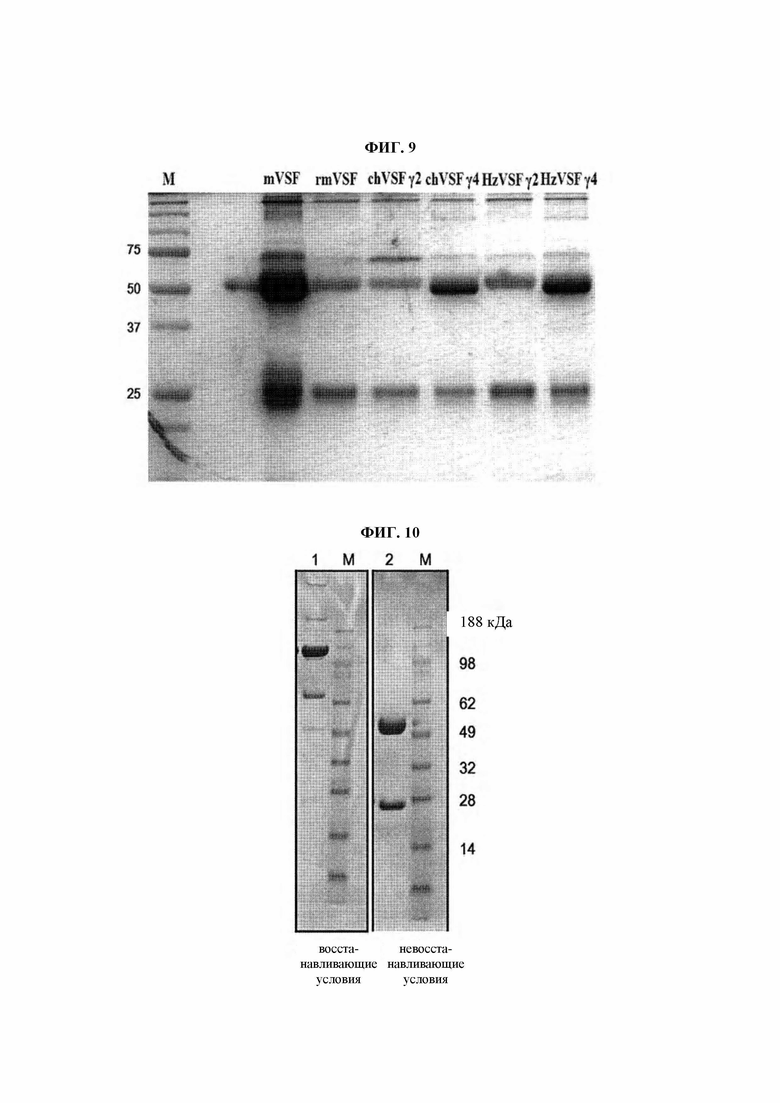
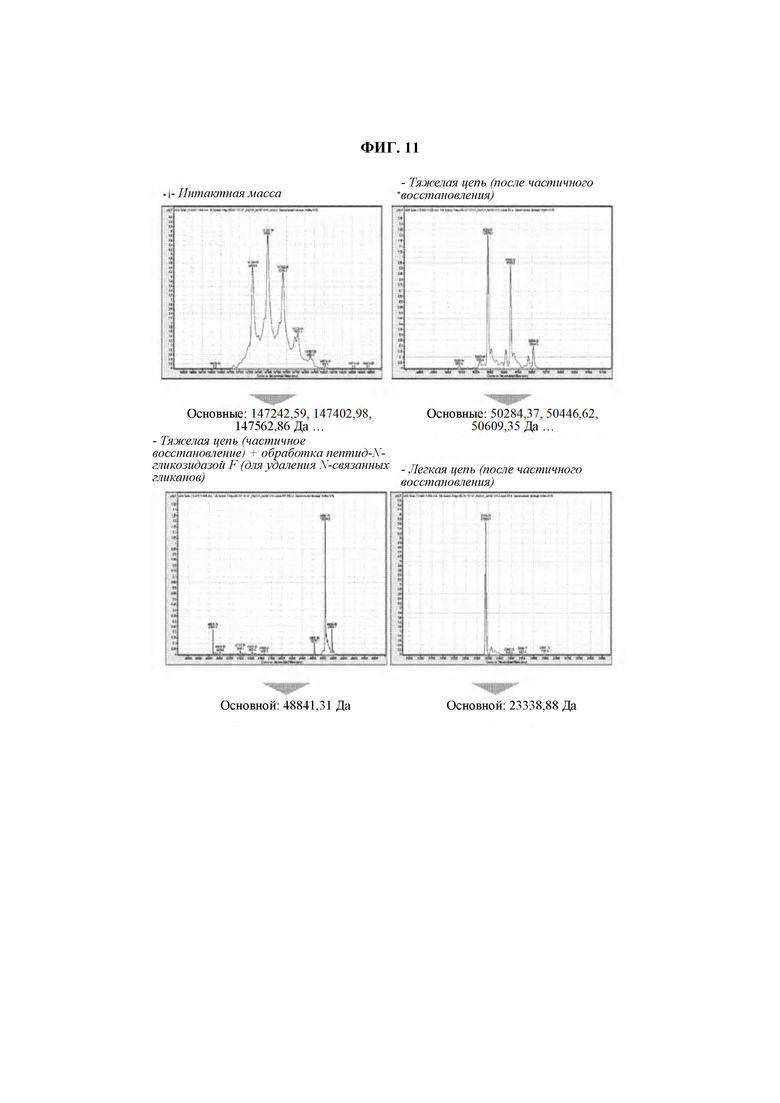
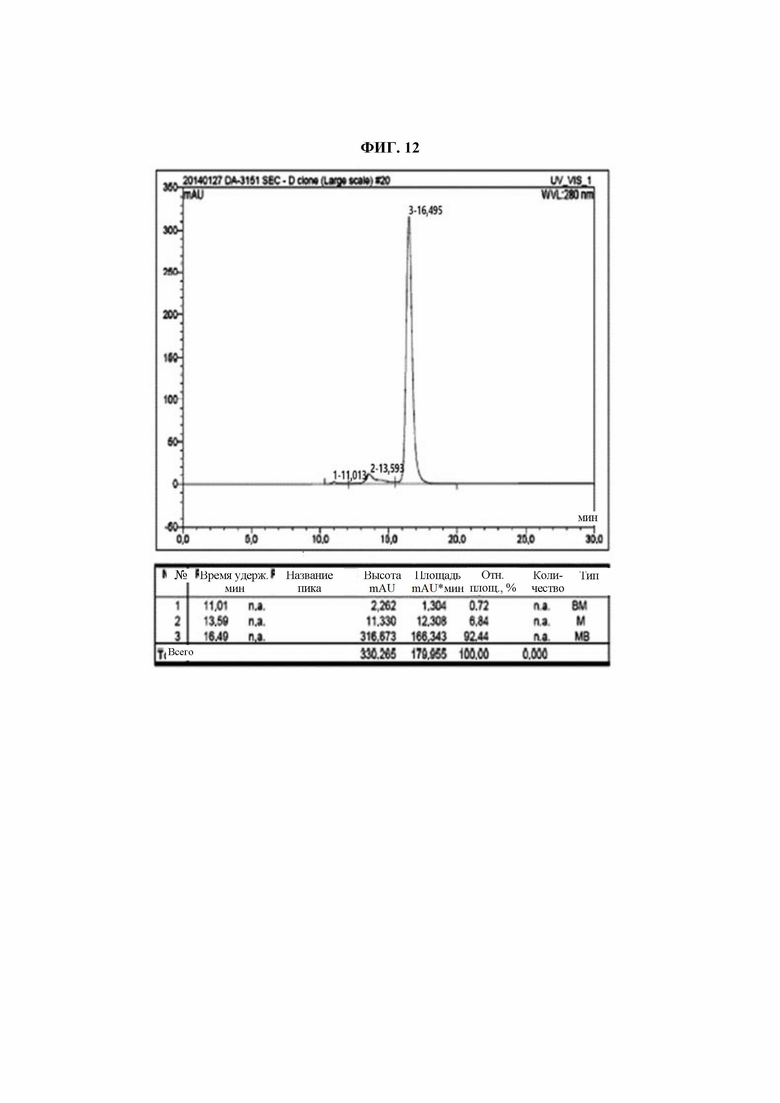
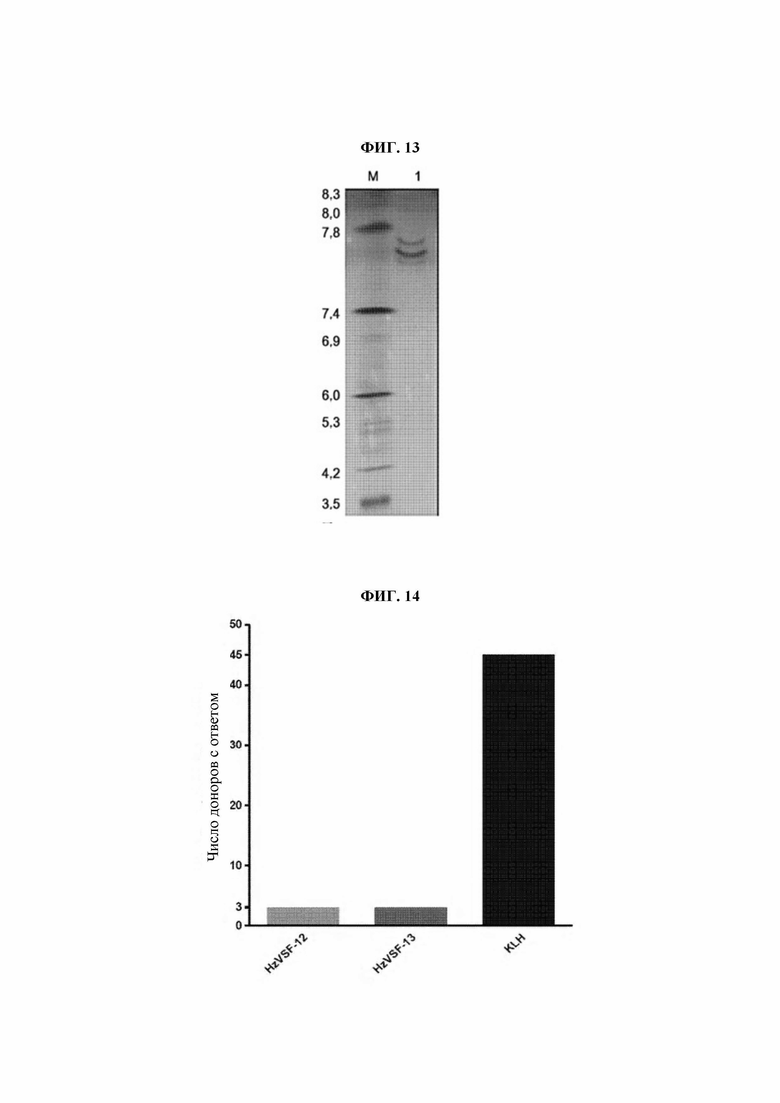
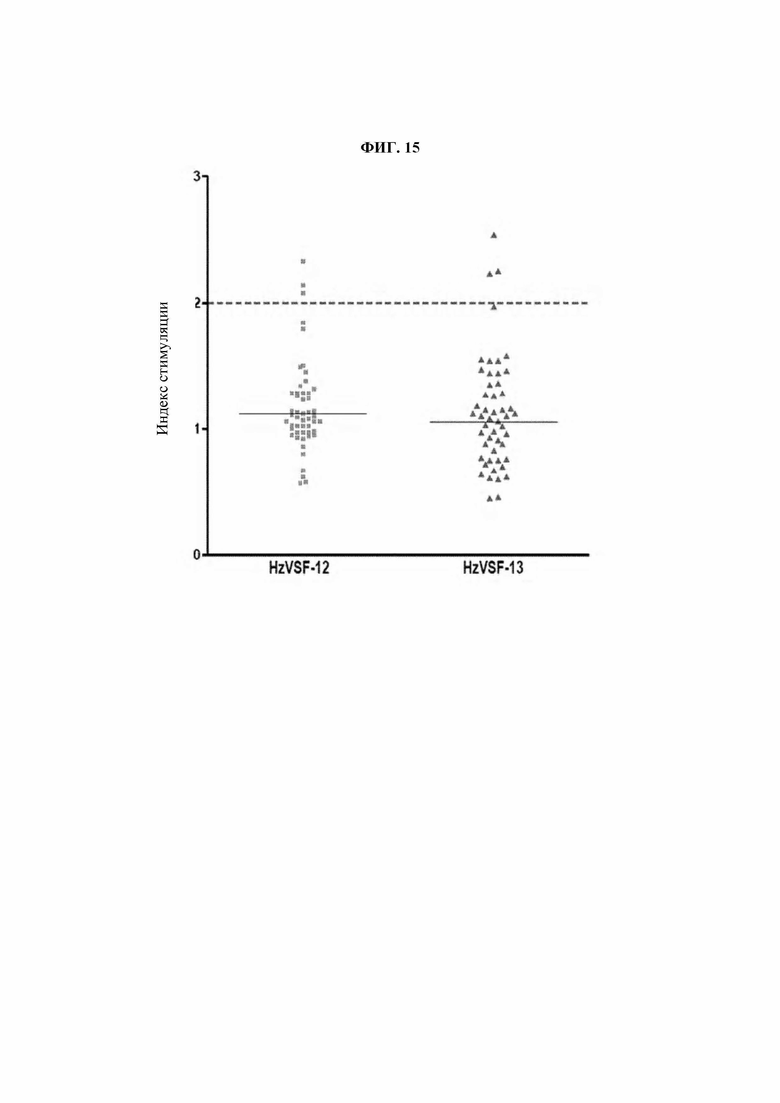
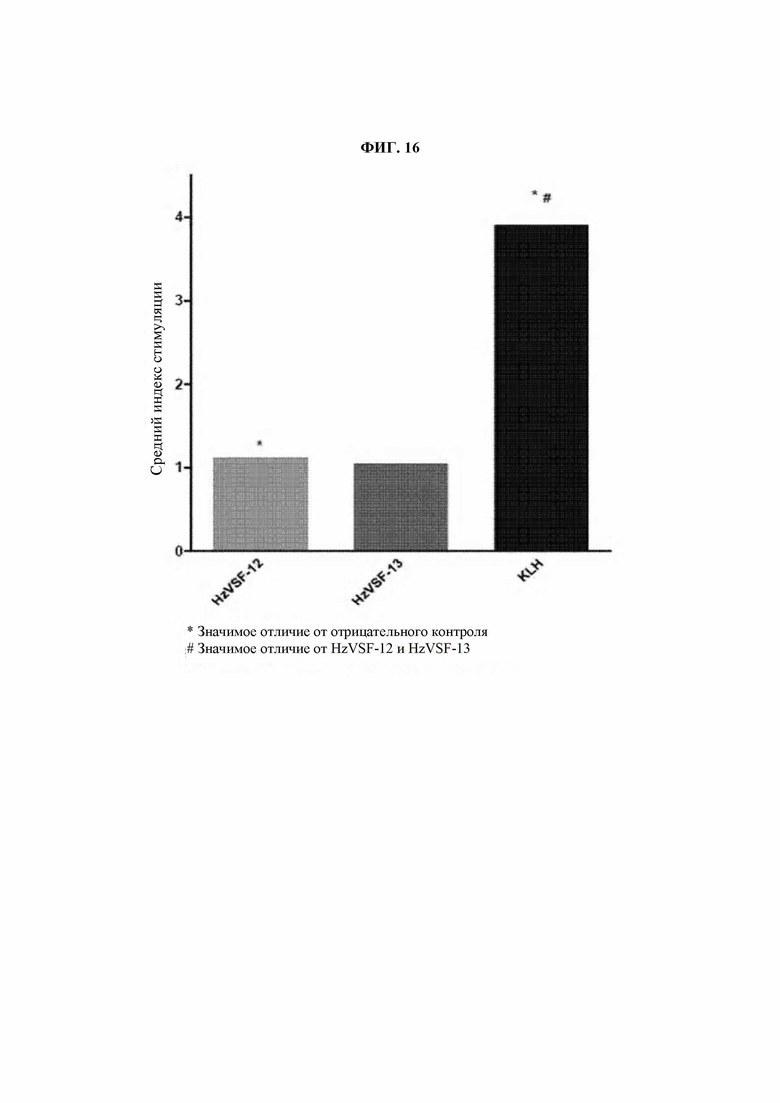

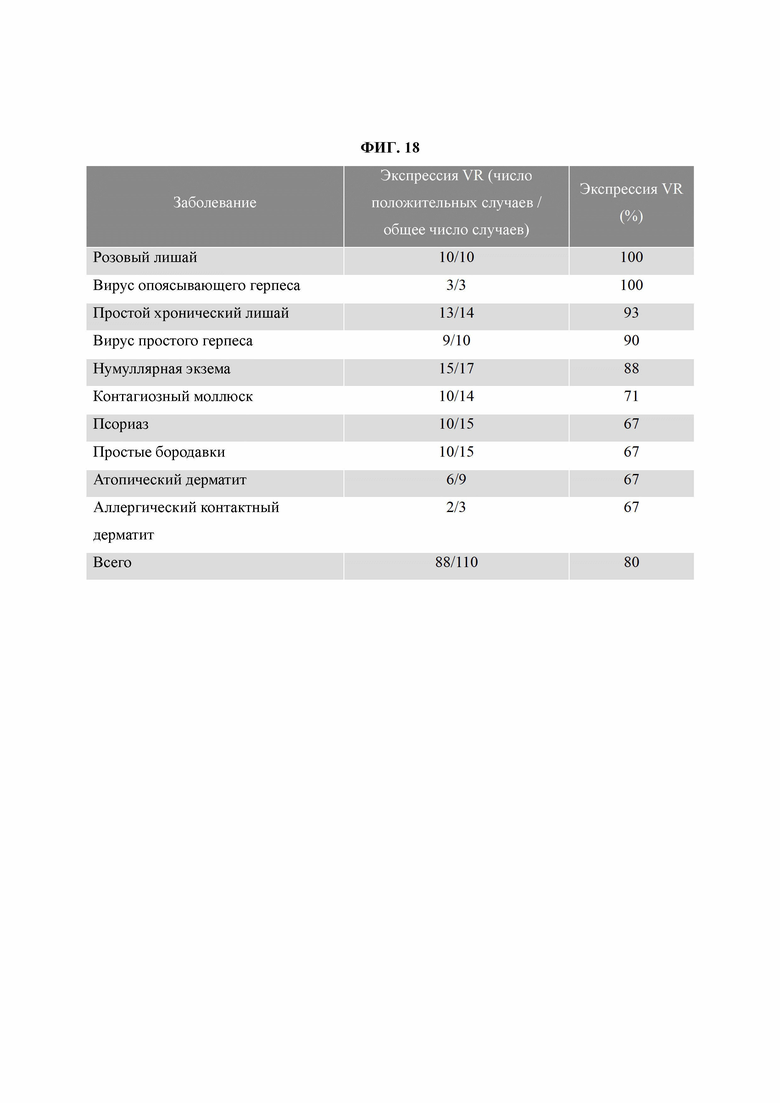
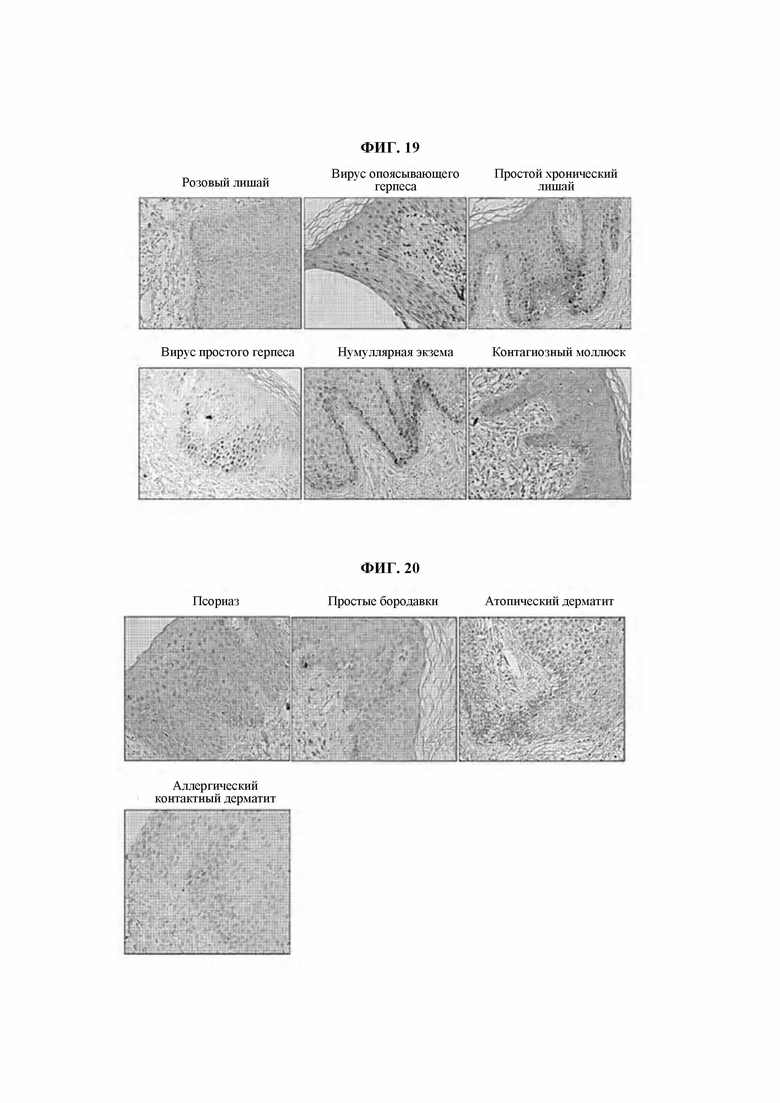
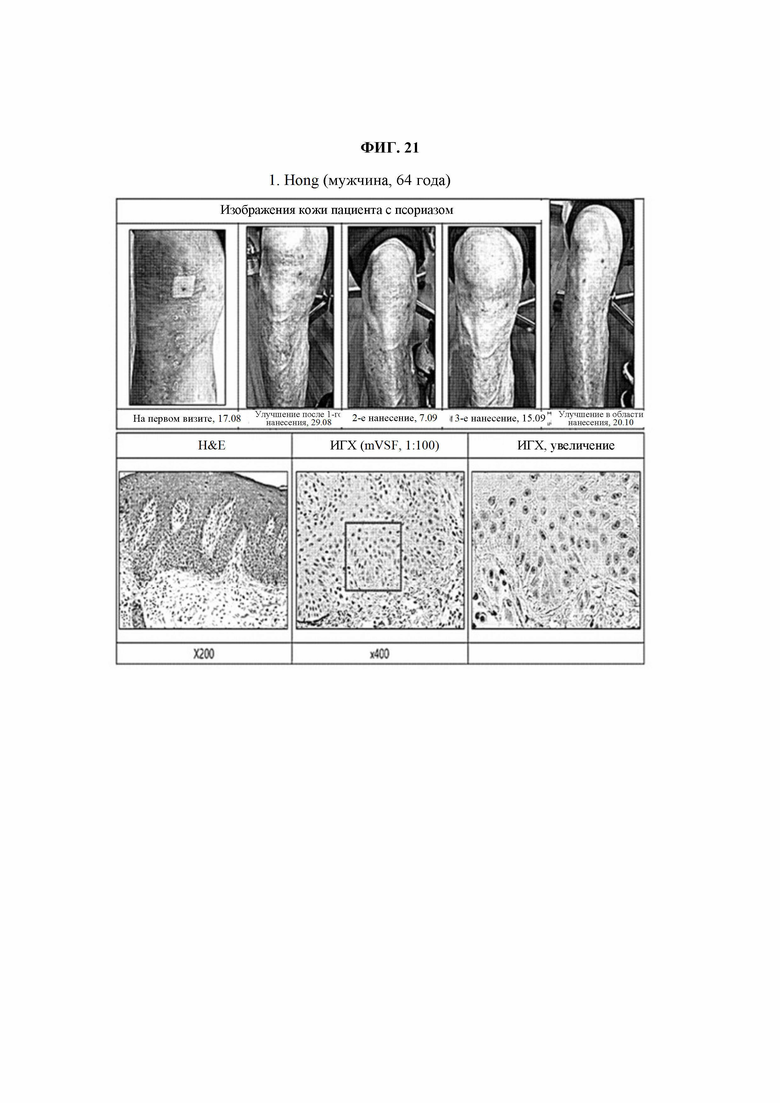
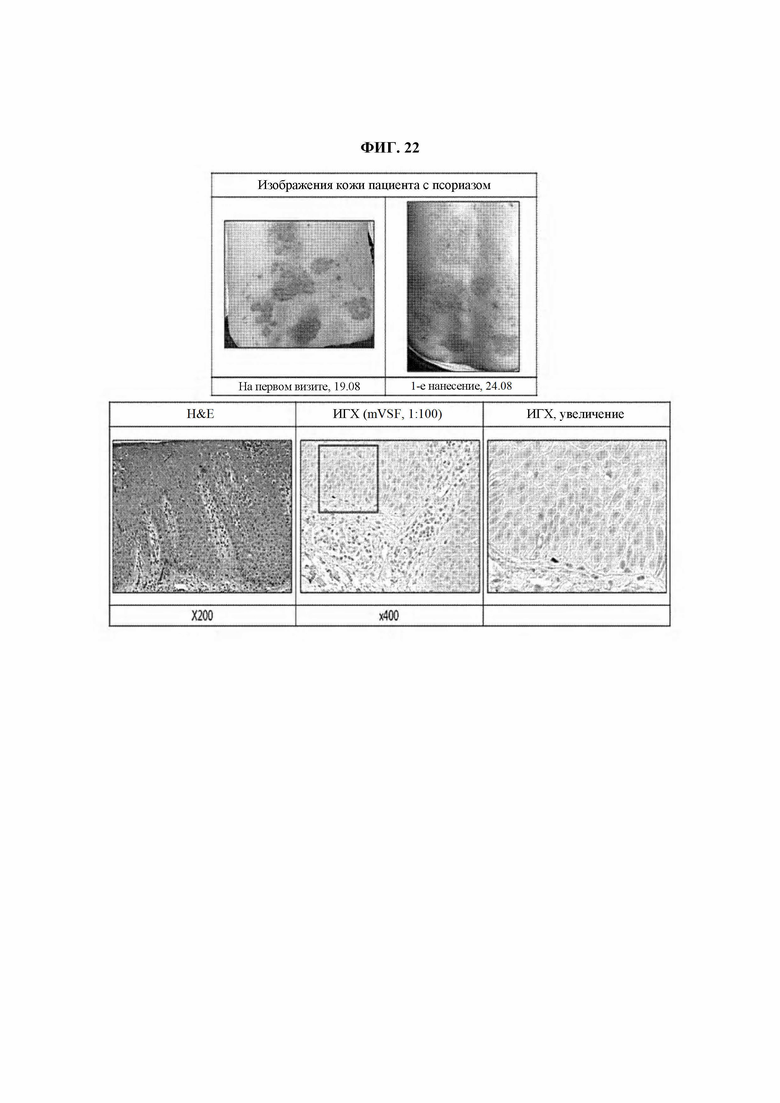
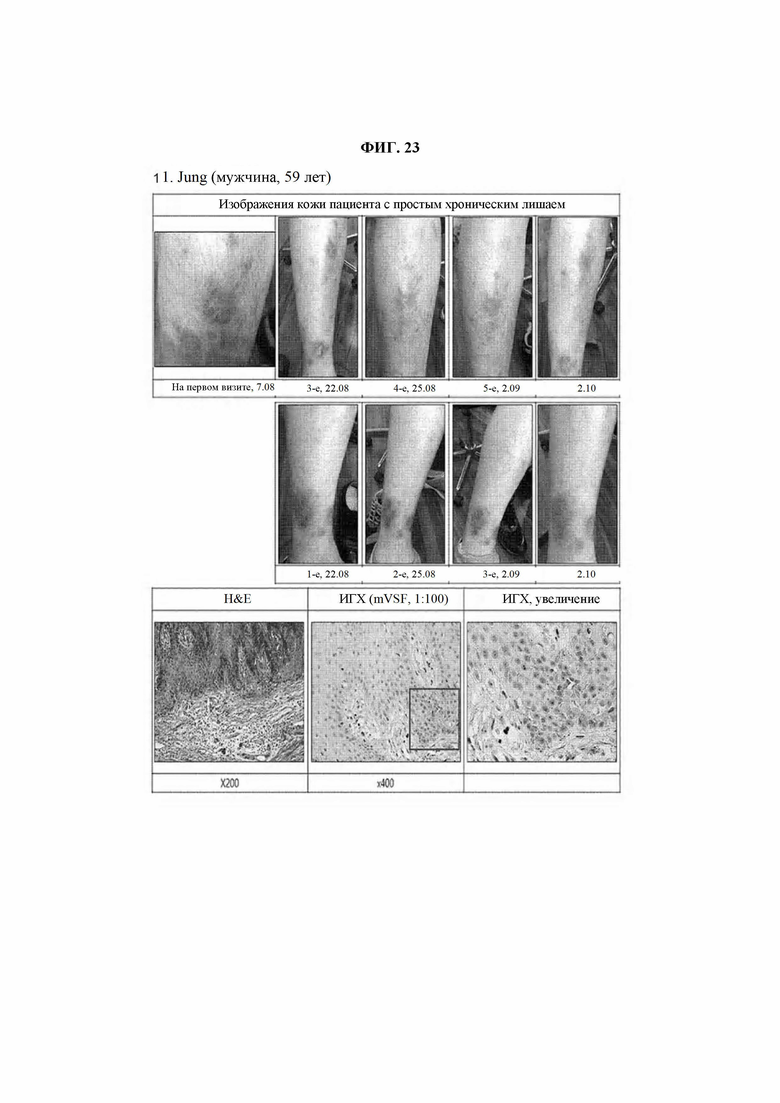
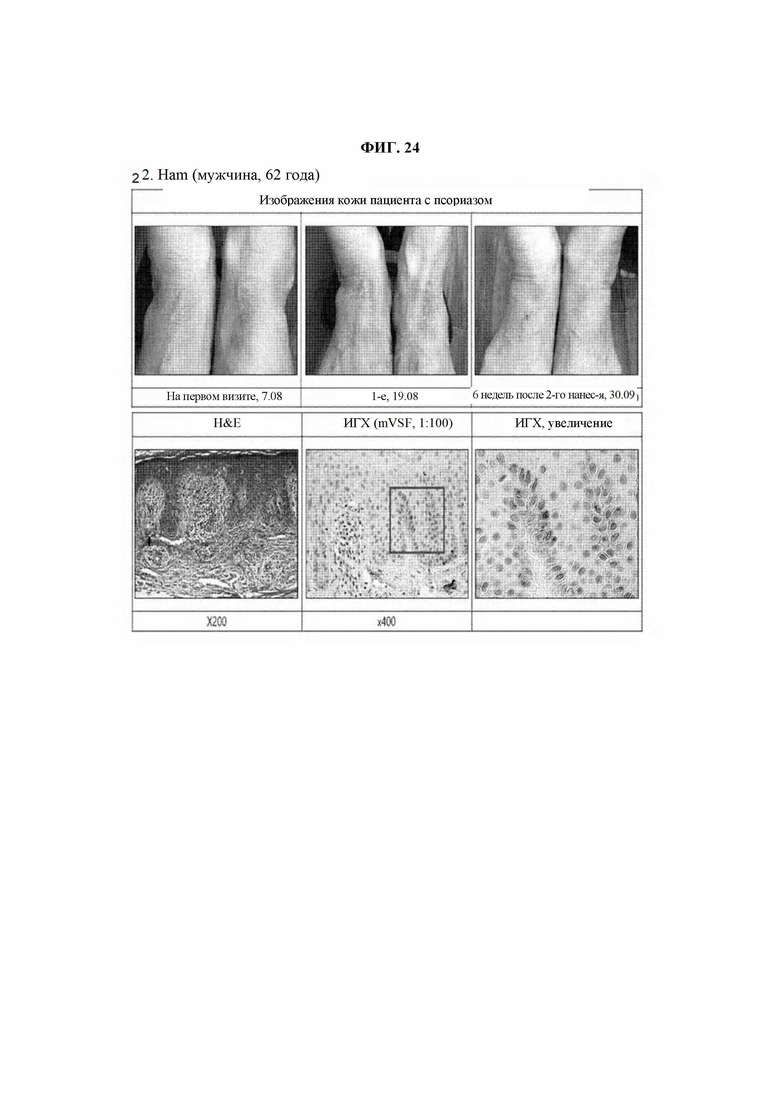
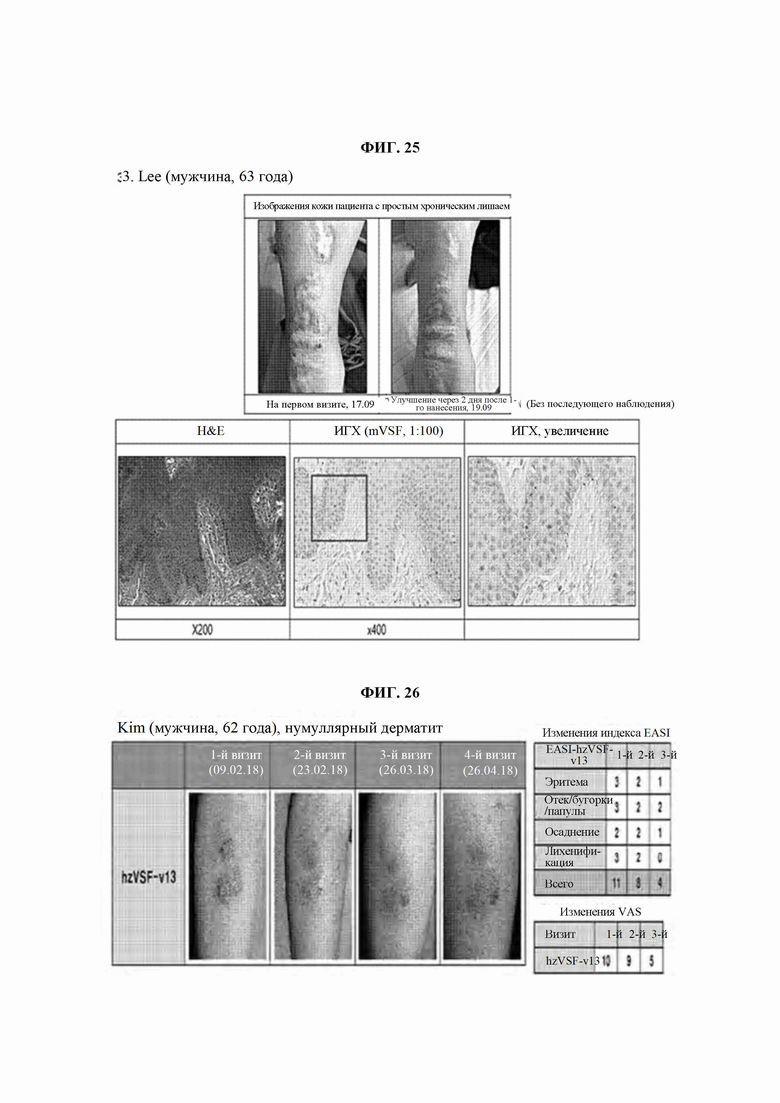
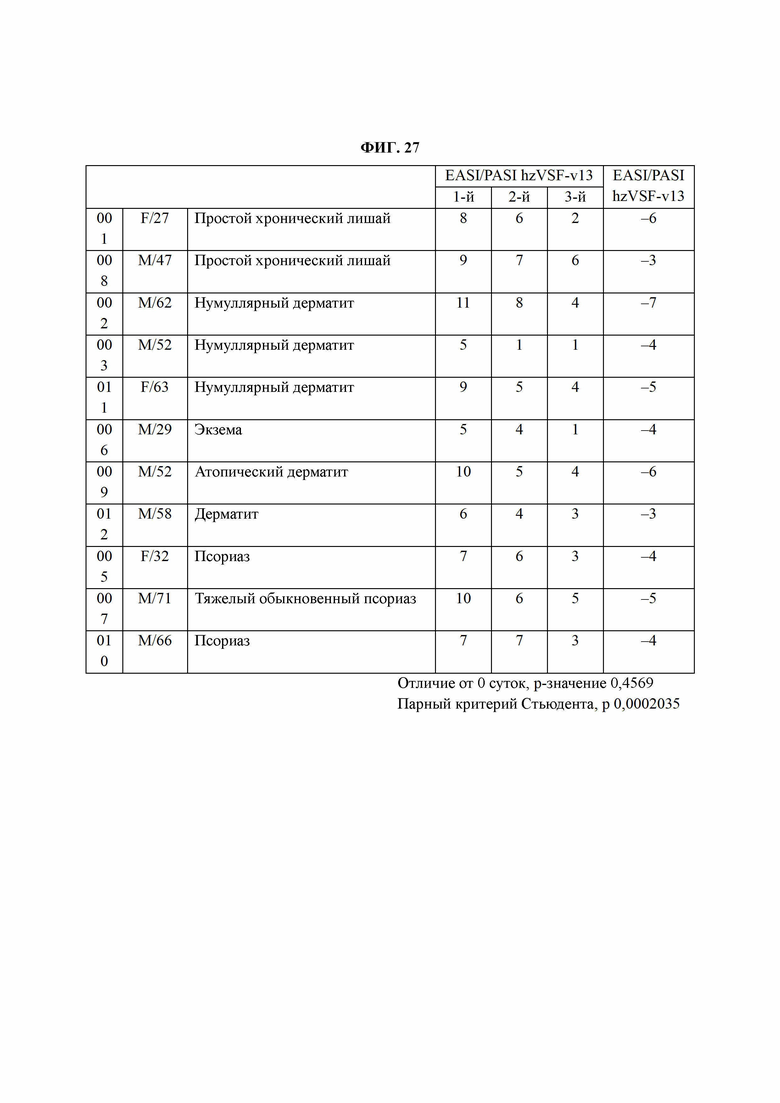
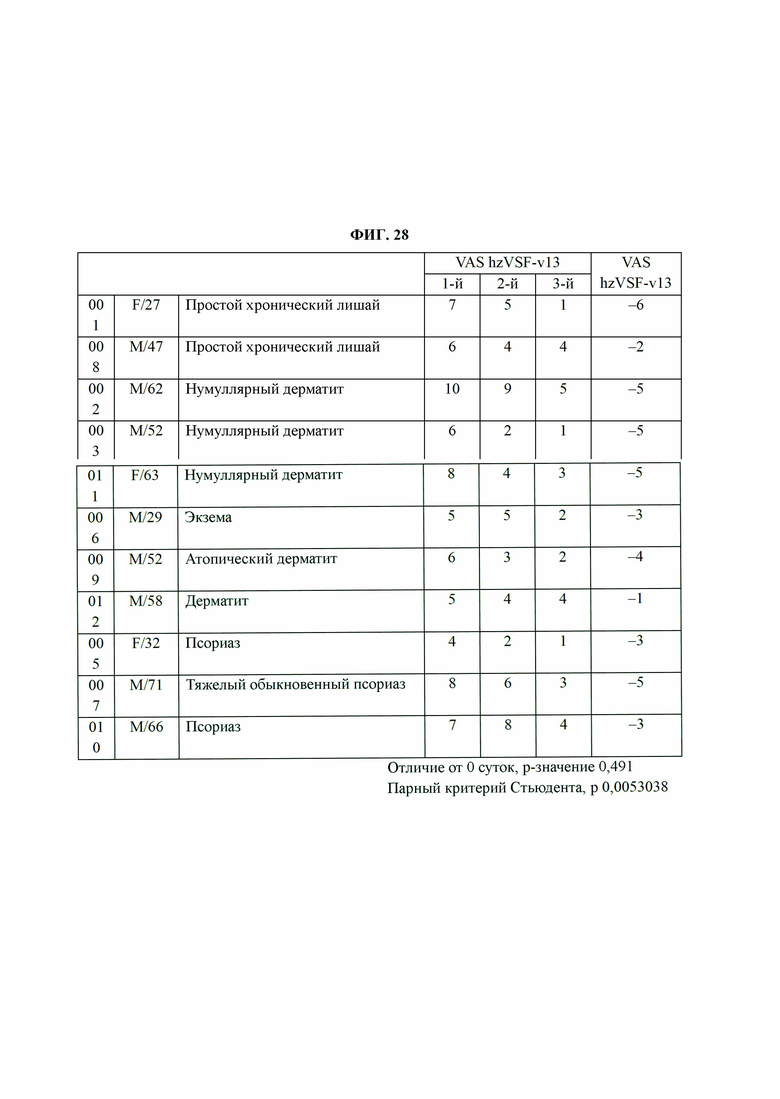
Настоящее изобретение относится к фармацевтической композиции для предупреждения или лечения заболеваний кожи, содержащая эффективное количество антитела или его фрагмента, специфично связывающихся с пептидом SEQ ID NO:1, и фармацевтически приемлемый носитель, где заболевание кожи представляет собой розовый лишай, опоясывающий герпес, простой хронический лишай, простой герпес, нумуллярную экзему, экзему, контагиозный моллюск, псориаз, простые бородавки, атопический дерматит или аллергический контактный дерматит, а также квазилекарственную композицию, препарат для накожного применения, способ предупреждения или лечения заболеваний кожи и применение вышеуказанной композиции для предупреждения или лечения заболеваний кожи, а также для изготовления лекарственного средства и для изготовления квазилекарственных средств или препаратов для накожного применения. 7 н. и 4 з.п. ф-лы, 28 ил., 8 табл., 8 пр.
1. Фармацевтическая композиция для предупреждения или лечения заболеваний кожи, содержащая в качестве активного ингредиента эффективное количество антитела или его фрагмента, специфично связывающихся с пептидом SEQ ID NO:1, и фармацевтически приемлемый носитель, где заболевание кожи представляет собой розовый лишай, опоясывающий герпес, простой хронический лишай, простой герпес, нумуллярную экзему, экзему, контагиозный моллюск, псориаз, простые бородавки, атопический дерматит или аллергический контактный дерматит.
2. Фармацевтическая композиция по п. 1, где антитело представляет собой мышиное антитело, химерное антитело или гуманизированное антитело.
3. Фармацевтическая композиция по п. 1, где фрагмент представляет собой Fab, Fab', F(ab')2, scFv, dsFv, ds-scFv, их димер, мини-тело, диатело, мультимер или фрагмент биспецифичного антитела.
4. Фармацевтическая композиция по п. 2, где гуманизированное антитело или фрагмент содержит: вариабельную область тяжелой цепи, содержащую CDR1 тяжелой цепи SEQ ID NO:2, CDR2 тяжелой цепи SEQ ID NO:3 или SEQ ID NO:14 и CDR3 тяжелой цепи SEQ ID NO:4 или SEQ ID NO:15; и вариабельную область легкой цепи, содержащую CDR1 легкой цепи SEQ ID NO:5, CDR2 легкой цепи SEQ ID NO:6, SEQ ID NO:16, SEQ ID NO:17 или SEQ ID NO:18 и CDR3 легкой цепи SEQ ID NO:7 или SEQ ID NO:19.
5. Фармацевтическая композиция по п. 4, где гуманизированное антитело или фрагмент дополнительно содержит: вариабельную область тяжелой цепи, содержащую каркасную область 1 (FR1) тяжелой цепи SEQ ID NO:20, FR2 тяжелой цепи SEQ ID NO:21, FR3 тяжелой цепи SEQ ID NO:22 или SEQ ID NO:28 и FR4 тяжелой цепи SEQ ID NO:23, и вариабельную область легкой цепи, содержащую FR1 легкой цепи SEQ ID NO:24, FR2 легкой цепи SEQ ID NO:25, FR3 легкой цепи SEQ ID NO:26 и FR4 легкой цепи SEQ ID NO:27.
6. Квазилекарственная композиция для предупреждения или облегчения заболеваний кожи, содержащая в качестве активного ингредиента эффективное количество антитела или его фрагмента, специфично связывающихся с пептидом SEQ ID NO:1, и фармацевтически приемлемый носитель, где заболевание кожи представляет собой розовый лишай, опоясывающий герпес, простой хронический лишай, простой герпес, нумуллярную экзему, экзему, контагиозный моллюск, псориаз, простые бородавки, атопический дерматит или аллергический контактный дерматит.
7. Препарат для накожного применения для предупреждения или облегчения заболеваний кожи, содержащий в качестве активного ингредиента эффективное количество антитела или его фрагмента, специфично связывающихся с пептидом SEQ ID NO:1, и фармацевтически приемлемый носитель, где заболевание кожи представляет собой розовый лишай, опоясывающий герпес, простой хронический лишай, простой герпес, нумуллярную экзему, экзему, контагиозный моллюск, псориаз, простые бородавки, атопический дерматит или аллергический контактный дерматит.
8. Способ предупреждения или лечения заболеваний кожи, включающий введение или нанесение композиции, содержащей в качестве активного ингредиента антитело или его фрагмент, специфично связывающиеся с пептидом SEQ ID NO:1, субъекту, где заболевание кожи представляет собой розовый лишай, опоясывающий герпес, простой хронический лишай, простой герпес, нумуллярную экзему, экзему, контагиозный моллюск, псориаз, простые бородавки, атопический дерматит или аллергический контактный дерматит.
9. Применение композиции для предупреждения или лечения заболеваний кожи, содержащей в качестве активного ингредиента антитело или его фрагмент, специфично связывающиеся с пептидом SEQ ID NO:1, где заболевание кожи представляет собой розовый лишай, опоясывающий герпес, простой хронический лишай, простой герпес, нумуллярную экзему, экзему, контагиозный моллюск, псориаз, простые бородавки, атопический дерматит или аллергический контактный дерматит.
10. Применение композиции, содержащей в качестве активного ингредиента антитело или его фрагмент, специфично связывающиеся с пептидом SEQ ID NO:1, для изготовления лекарственного средства для предупреждения или лечения заболеваний кожи, где заболевание кожи представляет собой розовый лишай, опоясывающий герпес, простой хронический лишай, простой герпес, нумуллярную экзему, экзему, контагиозный моллюск, псориаз, простые бородавки, атопический дерматит или аллергический контактный дерматит.
11. Применение композиции, содержащей в качестве активного ингредиента антитело или его фрагмент, специфично связывающиеся с пептидом SEQ ID NO:1, для изготовления квазилекарственных средств или препаратов для накожного применения для облегчения заболеваний кожи, где заболевание кожи представляет собой розовый лишай, опоясывающий герпес, простой хронический лишай, простой герпес, нумуллярную экзему, экзему, контагиозный моллюск, псориаз, простые бородавки, атопический дерматит или аллергический контактный дерматит.
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| GLADE C.P | |||
| et al., Epidermal cell DNA content and intermediate filaments keratin 10 and vimentin after treatment of psoriasis with calcipotriol cream once daily, twice daily and in combination with clobetasone 17‐butyrate cream or betamethasone 17‐valerate cream: a comparative flow cytometric study, British | |||
Авторы
Даты
2021-07-14—Публикация
2018-08-30—Подача