Область техники, к которой относится изобретение
Настоящее изобретение касается антител против лиганда-1 запрограммированной клеточной смерти (PD-L1) человека либо их антигенсвязывающих фрагментов, кодирующих таковые нуклеиновых кислот, векторов, содержащих эти нуклеиновые кислоты, клеток, трансформированных этими векторами, способа получения этих антител либо их антигенсвязывающих фрагментов и содержащих их композиций для профилактики или лечения рака или инфекционных заболеваний.
Уровень техники
Иммунная реакция клеток антиген-специфичных Т-лимфоцитов – очень сложный процесс, который подлежит тонкой регуляции. Прежде всего, активация Т-лимфоцитов начинается тогда, когда находящийся на поверхности Т-лимфоцитов Т-клеточный антигеновый рецептор (TCR) распознает главный комплекс гистосовместимости (MHC) у антиген-презентирующих клеток (APC), а у человека – антигены, связанные с молекулами HLA (лейкоцитарных антигенов человека) класса II. При этом для достаточной активации Т-лимфоцитов, наряду с распознаванием антигена, требуются костимуляторные сигналы, которые получаются, когда CD80, CD40 и т.д., экспрессированные в антиген-презентирующих клетках, одновременно связываются с CD28, CD40L и т.д., то есть с лигандами, находящимися на поверхности клеток Т-лимфоцитов. При этом активируется секреция цитокинов. Активация Т-лимфоцитов не происходит без передачи костимулирующих сигналов, хотя сам антиген и распознается при связывании с TCR-MHC/эпитопом.
Однако также активируются и коингибиторные сигналы, так что активированные Т-лимфоциты через некоторое время становятся неактивными. Это может предотвратить повреждение тканей и т.п. вследствие чрезмерной иммуностимуляции. Существует множество коингибиторных сигналов, к примеру, в CD80 и CD86 задействован антиген цитотоксических Т-лимфоцитов CTLA-4 и белок-1 запрограммированной смерти (PD-1) Т-лимфоцитов и антиген-презентирующих клеток и соответствующие им лиганды типа PD-L1 (лиганд-1 запрограммированной смерти). CTLA-4 функционирует, инактивируя наивные или запоминающие Т-лимфоциты путем связывания с лигандами, CD80 и CD86. PD-1 функционирует, регулируя функции T-лимфоцитов в периферических тканях через PD-L1 и PD-L2.
Иммунологическая функция человеческого организма заключается в распознавании антигенов и в то же время в регулировании общих функций Т-лимфоцитов через регуляцию этих костимулирующих и коингибирующих сигналов. Этот регуляторный механизм называется “контрольные точки иммунитета”. Иммунологическая функция у человека заключается в выявлении опухолеспецифичных неоантигенов, экспрессируемых при таких вариациях, как мутации, возникающие в опухолевых клетках, и тем самым в устранении опухолевых клеток или источников вирусной инфекции.
С другой стороны, чтобы избежать таких иммунных атак, некоторые опухолевые клетки ингибируют иммунологические функции путем изменения микроокружения опухоли или иммунного избегания посредством иммунотолерантности Т-клеток или иммуноредактирования.
Одна из стратегий иммунного избегания заключается в ингибировании функций опухолеспецифичных Т-лимфоцитов посредством изменения функций контрольных точек иммунитета. То есть атака опухолеспецифичных клеток Т-лимфоцитов устраняется путем активации таких ингибиторных точек иммунитета в опухолевых клетках. При этом активность и действие ингибированных опухолеспецифичных клеток Т-лимфоцитов можно улучшить путем ингибирования их функций с помощью моноклональных антител против PD-1 или его лиганда PD-L1 с тем, чтобы получить противораковые эффекты.
На этом техническом фоне авторы настоящего изобретения приложили усилия для разработки антител, специфически связывающихся с PD-L1. В результате они разработали антитела против PD-L1, которые связываются с высоким сродством с PD-L1, и обнаружили, что такие антитела против PD-L1 могут служить в качестве требуемых иммунологических противораковых средств или терапевтических средств от инфекционных заболеваний при ингибировании образования комплекса PD-1/PD-L1, и тем самым совершили настоящее изобретение.
Сущность изобретения
Техническая проблема
Итак, одной из целей настоящего изобретения является получение новых антител к PD-L1 либо их антигенсвязывающих фрагментов.
Другой целью настоящего изобретения является получение нуклеиновых кислот, кодирующих эти антитела либо их антигенсвязывающие фрагменты.
Следующей целью настоящего изобретения является получение векторов, содержащих эти нуклеиновые кислоты, клеток, трансформированных этими векторами, и способа получения таковых.
Следующей целью настоящего изобретения является получение композиций для профилактики или лечения рака либо инфекционных заболеваний, содержащих эти антитела либо их антигенсвязывающие фрагменты.
Техническое решение
В соответствии с настоящим изобретением, вышеуказанные и другие цели могут быть достигнуты путем получения антитела, связывающегося с PD-L1, либо его антиген-связывающего фрагмента, содержащее: вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 1-7, CDR2 тяжелой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 8-15, и CDR3 тяжелой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 16-25; и вариабельную область легкой цепи, включающую CDR1 легкой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 88-102, CDR2 легкой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 103-119, и CDR3 легкой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 120-144.
В соответствии с другим аспектом настоящего изобретения предусмотрена нуклеиновая кислота, кодирующая данное антитело или антигенсвязывающий фрагмент.
В соответствии с другим аспектом настоящего изобретения предусмотрен экспрессионный вектор, содержащий данную нуклеиновую кислоту.
В соответствии с другим аспектом настоящего изобретения предусмотрена клетка, трансформированная данным экспрессионным вектором.
В соответствии с другим аспектом настоящего изобретения предусмотрен способ получения антитела либо его антигенсвязывающего фрагмента, включающий (a) культивирование клетки и (b) извлечение антитела либо его антигенсвязывающего фрагмента из культивированной клетки.
В соответствии с другим аспектом настоящего изобретения предусмотрена композиция для профилактики или лечения рака либо инфекционных заболеваний, содержащая в качестве активного ингредиента данное антитела либо его антигенсвязывающий фрагмент.
Краткое описание фигур
Вышеуказанные и другие цели, особенности и другие преимущества настоящего изобретения станут более понятными из следующего подробного описания в сочетании с прилагаемыми чертежами.
На фиг. 1 представлена схематическая диаграмма экспрессирующего PD-L1 вектора.
На фиг. 2 представлены результаты по очистке белка PD-L1.
На фиг. 2A представлены результаты по идентификации белка в отношении PD-L1-hFc в 10% геле SDS-PAGE в восстановительных (RE) и невосстановительных (NR) условиях.
На фиг. 2B представлены результаты по SEC-HPLC на G-3000 SWXL при скорости потока 1 мл/мин с использованием PBS в качестве рабочего растворителя.
На фиг. 2C представлены результаты по идентификации белка в отношении PD-L1-mFc в 10% геле SDS-PAGE в восстановительных (RE) и невосстановительных (NR) условиях.
На фиг. 2D представлены результаты по SEC-HPLC на G-3000 SWXL при скорости потока 1 мл/мин с использованием PBS в качестве рабочего растворителя.
На фиг. 3 представлены результаты по повышению способности к связыванию с антигеном PD-L1 в зависимости от количества раундов пэннинга.
На фиг. 4 представлены результаты ELISA по измерению способности монофагов к связыванию с высоким сродством только с PD-L1-His.
На фиг. 5 представлены результаты анализа SDS-PAGE для идентификации выбранных антител к PD-L1.
На фиг. 6 представлены результаты по оценке эффективности антител к PD-L1 in vitro.
На фиг. 7 представлены результаты по оценке зависимой от концентрации эффективности антител к PD-L1 in vitro.
На фиг. 8 представлены результаты по измерению способности антител к PD-L1 связываться на клетках, гиперэкспрессирующих PD-L1.
На фиг. 9 представлены результаты по измерению кинетики связывания между PD-L1-hFc и антителом 16E12 к PD-L1.
На фиг. 10 представлены результаты скрининга по оптимизации моноклонов.
На фиг. 11 представлены результаты по оценке эффективности in vitro в отношении антител к PD-L1 по настоящему изобретению.
На фиг. 12 представлены результаты по оценке зависимой от концентрации эффективности in vitro антител к PD-L1 по настоящему изобретению.
На фиг. 13 представлены результаты по измерению способности антител к связыванию на клетках, гиперэкспрессирующих PD-L1.
На фиг. 14 представлены результаты по идентификации методом ферментного иммуноанализа в отношении ингибирующей активности выбранных антител по предотвращению образования комплекса PD-1/PD-L2.
На фиг. 15 представлены результаты по измерению кинетики связывания между PD-L1-hFc и антителами 16E12 и 4F5 к PD-L1.
На фиг. 16 представлены результаты по измерению связывания между мутантными белками PD-L1 и моноклональными антителами.
На фиг. 17 представлены результаты по идентификации моноклональных антител к PD-L1 в отношении повышения активности при гетерогенной MLR (реакции смешанных лимфоцитов).
На фиг. 18 представлены результаты по оценке эффективности выбранных моноклональных антител к PD-L1 на модели рака у сингенных животных.
На фиг. 19 представлены результаты по идентификации в отношении связывания антител против PD-L1 по настоящему изобретению с PD-L2.
Раскрытие сущности изобретения
Если не указано иначе, все используемые здесь технические и научные термины имеют такие же значения, как те, что известны специалистам в той области, к которой относится настоящее изобретение. В общем, используемая здесь номенклатура хорошо известна в данной области, обычно она и применяется.
В одном аспекте настоящее изобретение направлено на антитело, связывающееся с PD-L1, либо его антигенсвязывающим фрагментом, содержащее: вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 1-7, CDR2 тяжелой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 8-15, и CDR3 тяжелой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 16-25; и вариабельную область легкой цепи, включающую CDR1 легкой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 88-102, CDR2 легкой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 103-119, и CDR3 легкой цепи, последовательность которого на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 120-144.
В настоящем изобретении термин “PD-L1” означает лиганд для иммуносупрессивного рецептора “белка-1 запрограммированной смерти (PD-1)”, который преимущественно экспрессируется в активированных T- и B-клетках, которые могут отрицательно регулировать сигнальный путь антигеновых рецепторов. Лиганды (PD-L1 и PD-L2) для PD-1 могут быть экспрессироваться конститутивно или могут вырабатываться в различных типах клеток, включая клетки негематопоэтических тканей и различных типов опухолей. PD-L1 экспрессируется в B-клетках, T-клетках, клетках костного мозга и дендритных клетках (DC), а также в таких нелимфатических органах, как периферические клетки, псевдососудистые эндотелиальные клетки и сердце, легкие и др. Напротив, PD-L2 встречается только в макрофагах и дендритных клетках. Профиль экспрессии лигандов PD-1 свидетельствует о роли PD-1 в поддержании периферической толерантности, что может способствовать регуляции аутореактивных T-клеточных и B-клеточных ответов на периферии. Оба лиганда представляют собой трансмембранные рецепторы типа I, которые содержат домены типа IgV и IgC во внеклеточной части. Оба лиганда включают в себя короткий цитоплазматический участок, содержащий неизвестный сигнальный мотив.
В ряде исследований было показано, что взаимодействие между PD-1 и его лигандами ингибирует пролиферацию лимфоцитов in vitro и in vivo. Известно, что нарушение взаимодействия PD-1/PD-L1 усиливает пролиферацию Т-клеток и продукцию цитокинов, а также блокирует прохождение клеточного цикла. Блокирование взаимодействия PD-1/PD-L1 может привести к улучшению опухолеспецифичного T-клеточного иммунитета, способствуя тем самым очистке от опухолевых клеток иммунной системой. Кроме того, при хронической ВИЧ-инфекции нарушаются функции ВИЧ-специфичных Т-клеток CD8+, у которых снижается способность к продукции цитокинов и эффекторных молекул и уменьшается способность к пролиферации, причем у ВИЧ-специфичных T-клеток CD8+ сильно экспрессируется PD-1, который может улучшать активность Т-клеток или противовирусные иммунные реакции путем усиления способности к пролиферации ВИЧ-специфичных Т-клеток и способности к продукции цитокинов в ответ на пептидные стимулы ВИЧ посредством блокировки взаимодействия PD-1/PD-L1.
В настоящем изобретении термин “антитело” означает антитело против PD-L1, которое специфически связывается с PD-L1. В рамки настоящего изобретения входят не только полные антитела, специфически связывающиеся с PD-L1, но также и антигенсвязывающие фрагменты молекул антител.
Полное антитело означает структуру, содержащую две полноразмерные легкие цепи и две полноразмерные тяжелые цепи, причем каждая легкая цепь связана с соответствующей тяжелой цепью дисульфидной связью. Константная область тяжелой цепи бывает типа гамма (γ), мю (μ), альфа (α), дельта (δ) или эпсилон (ε) и подразделяется на гамма-1 (γ1), гамма-2 (γ2), гамма-3 (γ3), гамма-4 (γ4), альфа-1 (α1) и альфа-2 (α2). Константная область легкой цепи бывает типа каппа (κ) и лямбда (λ).
Антигенсвязывающий фрагмент антитела или же фрагмент антитела означает такой фрагмент, который по меньшей мере обладает способностью к связыванию антигена, и охватывает Fab, F(ab′), F(ab′)2 и Fv. Из всех фрагментов антител Fab означает структуру, включающую вариабельную область каждой из тяжелых и легких цепей, константный домен легкой цепи и первый константный домен (CH1) тяжелой цепи, каждый из которых содержит один антигенсвязывающий сайт. Fab′ отличается от Fab тем, что он дополнительно включает шарнирный участок, содержащий по меньшей мере один остаток цистеина на С-конце домена СН1 тяжелой цепи. F(ab′)2 образуется по дисульфидной связи между остатками цистеина в шарнирном участке Fab′. Fv – минимальный фрагмент антитела, содержащий только вариабельную область тяжелой цепи и вариабельную область легкой цепи, а рекомбинантная технология получения Fv изложена в таких международных публикациях PCT, как WO 88/01649, WO 88/06630, WO 88/07085, WO 88/07086 и WO 88/09344. Двухцепочечный Fv представляет собой фрагмент, в котором вариабельная область тяжелой цепи и вариабельная область легкой цепи связаны нековалентной связью, а одноцепочечный Fv представляет собой фрагмент, в котором вариабельная область тяжелой цепи и вариабельная область легкой цепи обычно связаны ковалентной связью через пептидный линкер или непосредственно связаны на С-конце, образуя димероподобную структуру типа двухцепочечного Fv. Такие фрагменты антител могут быть получены с помощью протеаз (например, Fabs могут быть получены путем рестрикционного расщепления всего антитела папаином, а F(ab′)2-фрагмент может быть получен путем рестрикционного расщепления всего антитела пепсином), а также могут быть получены методами генетической рекомбинации.
В одном воплощении антитела по настоящему изобретению представлены в виде Fv (к примеру, scFv), Fab или полного антитела. Кроме того, константная область тяжелой цепи может быть выбрана из изотипов, состоящих из гамма (γ), мю (μ), альфа (α), дельта (δ) или эпсилон (ε). Например, константная область может быть типа гамма-1 (IgG1), гамма-3 (IgG3) или гамма-4 (IgG4). Константная область легкой цепи может быть типа каппа или лямбда.
В настоящем изобретении термин “тяжелая цепь” охватывает как полноразмерные тяжелые цепи, которые включают вариабельный домен (VH), аминокислотная последовательность которого содержит достаточную последовательность вариабельной области для придания специфичности к антигену, и три константных домена (СН1, СН2 и СН3), так и их фрагменты. В настоящем изобретении термин “легкая цепь” охватывает как полноразмерные легкие цепи, которые включают вариабельный домен (VL), аминокислотная последовательность которого содержит достаточную последовательность вариабельной области для придания специфичности к антигену, и константный домен (CL), так и их фрагменты.
Антитела по настоящему изобретению включают, без ограничения, моноклональные антитела, мультиспецифичные антитела, человеческие антитела, гуманизованные антитела, химерные антитела, короткоцепочечные Fv (scFv), короткоцепочечные антитела, Fab-фрагменты, F(ab′)-фрагменты, связанные дисульфидными связями Fv (sdFVs), антиидиотипические (анти-Id) антитела или же эпитоп-связывающие фрагменты таких антител и т.п.
Моноклональные антитела означают такие антитела, которые получены из популяции практически однородных антител, за исключением возможных природных мутаций, то есть каждое антитело, составляющее данную популяцию, может присутствовать в незначительном количестве. Моноклональные антитела очень специфичны и вырабатываются против одного антигенного сайта. В отличие от препаратов обычных (поликлональных) антител, которые обычно содержат разные антитела, направленные на различные детерминанты (эпитопы), каждое моноклональное антитело направлено на одну детерминанту данного антигена.
Термин “эпитоп” означает белковую детерминанту, с которой антитело может специфически связываться. Эпитоп обычно состоит из химически активных групп на поверхности молекулы, к примеру, аминокислот или боковых цепей сахаров, и обычно обладают специфическими характеристиками трехмерной структуры, а также специфическими характеристиками заряда. Стерические и нестерические эпитопы отличаются друг от друга тем, что связывание со стерическими эпитопами теряется в присутствии денатурирующих растворителей, а связывание с нестерическими эпитопами – нет.
Нечеловеческие (например, мышиные) антитела “гуманизованной” формы представляют собой химерные антитела, содержащие минимальную последовательность, происходящую не из иммуноглобулина человека. В большинстве случаев гуманизованные антитела представляют собой иммуноглобулины человека (антитела-реципиенты), у которых остатки из гипервариабельной области реципиента заменены на остатки из гипервариабельной области другого вида, чем человек (антитела-донора), как-то мыши, крысы, кролика или приматов, отличных от человека, обладающие требуемой специфичностью, сродством и способностью.
Термин “человеческое антитело” обозначает молекулы, происходящие из иммуноглобулина человека, у которых все аминокислотные последовательности, составляющие антитело, включая определяющие комплементарность участки и структурные участки, состоят из иммуноглобулина человека.
Некоторые части тяжелой цепи и/или легкой цепи идентичны или гомологичны соответствующим последовательностям у антител, происходящих из определенного вида или принадлежащих к определенному классу или подклассу антител, тогда как остальные части включают “химерные” антитела (иммуноглобулины), которые идентичны или гомологичны соответствующим последовательностям у антител, происходящих из другого вида или принадлежащих к другому классу или подклассу антител, а также фрагменты таких антител, проявляющие требуемую биологическую активность.
В настоящем изобретении термин “вариабельный домен антитела” относится к областям легкой и тяжелой цепи молекул антител, включающим аминокислотные последовательности определяющих комплементарность участков (CDR; т.е. CDR1, CDR2 и CDR3) и каркасных участков (FR). VH означает вариабельный домен тяжелой цепи. VL означает вариабельный домен легкой цепи.
Термин “определяющий комплементарность участок” (CDR; т.е. CDR1, CDR2 и CDR3) относится к таким аминокислотным остаткам вариабельного домена антител, которые необходимы для связывания антигена. Каждый вариабельный домен обычно содержит три участка CDR, обозначаемые как CDR1, CDR2 и CDR3.
В настоящем изобретении антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты содержат: вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи, выбранный из группы, состоящей из SEQ ID NOs: 1-7, CDR2 тяжелой цепи, выбранный из группы, состоящей из SEQ ID NOs: 8-15, и CDR3 тяжелой цепи, выбранный из группы, состоящей из SEQ ID NOs: 16-25; и вариабельную область легкой цепи, включающую CDR1 легкой цепи, выбранный из группы, состоящей из SEQ ID NOs: 88-102, CDR2 легкой цепи, выбранный из группы, состоящей из SEQ ID NOs: 103-119, и CDR3 легкой цепи, выбранный из группы, состоящей из SEQ ID NOs: 120-144.
В частности, антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты по настоящему изобретению содержат:
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 1, CDR2 тяжелой цепи по SEQ ID NO: 8 и CDR3 тяжелой цепи по SEQ ID NO: 16;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 18;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 3, CDR2 тяжелой цепи по SEQ ID NO: 10 и CDR3 тяжелой цепи по SEQ ID NO: 19;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 4, CDR2 тяжелой цепи по SEQ ID NO: 11 и CDR3 тяжелой цепи по SEQ ID NO: 20;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 5, CDR2 тяжелой цепи по SEQ ID NO: 12 и CDR3 тяжелой цепи по SEQ ID NO: 21;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 6, CDR2 тяжелой цепи по SEQ ID NO: 13 и CDR3 тяжелой цепи по SEQ ID NO: 22;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 23;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 7, CDR2 тяжелой цепи по SEQ ID NO: 14 и CDR3 тяжелой цепи по SEQ ID NO: 24;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 15 и CDR3 тяжелой цепи по SEQ ID NO: 25; или
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17.
Кроме того, антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты содержат:
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 88, CDR2 легкой цепи по SEQ ID NO: 103 и CDR3 легкой цепи по SEQ ID NO: 120;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 121;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 90, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 122;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 91, CDR2 легкой цепи по SEQ ID NO: 106 и CDR3 легкой цепи по SEQ ID NO: 123;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 107 и CDR3 легкой цепи по SEQ ID NO: 124;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 92, CDR2 легкой цепи по SEQ ID NO: 108 и CDR3 легкой цепи по SEQ ID NO: 122;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 93, CDR2 легкой цепи по SEQ ID NO: 109 и CDR3 легкой цепи по SEQ ID NO: 125;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 94, CDR2 легкой цепи по SEQ ID NO: 110 и CDR3 легкой цепи по SEQ ID NO: 126;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 95, CDR2 легкой цепи по SEQ ID NO: 111 и CDR3 легкой цепи по SEQ ID NO: 127;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 96, CDR2 легкой цепи по SEQ ID NO: 112 и CDR3 легкой цепи по SEQ ID NO: 128;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 108 и CDR3 легкой цепи по SEQ ID NO: 129;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 130;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 113 и CDR3 легкой цепи по SEQ ID NO: 131;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 97, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 132;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 133;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 97, CDR2 легкой цепи по SEQ ID NO: 114 и CDR3 легкой цепи по SEQ ID NO: 134;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 92, CDR2 легкой цепи по SEQ ID NO: 115 и CDR3 легкой цепи по SEQ ID NO: 135;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 98, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 130;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 116 и CDR3 легкой цепи по SEQ ID NO: 121;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 108 и CDR3 легкой цепи по SEQ ID NO: 136;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 99, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 137;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 117 и CDR3 легкой цепи по SEQ ID NO: 138;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 118 и CDR3 легкой цепи по SEQ ID NO: 133;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 119 и CDR3 легкой цепи по SEQ ID NO: 139;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 100, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 140;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 108 и CDR3 легкой цепи по SEQ ID NO: 141;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 139;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 142;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 143;
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 101, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 141; или
вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 102, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 144.
В одном воплощении настоящего изобретения антитела либо их антигенсвязывающие фрагменты по настоящему изобретению могут содержать следующие вариабельные области тяжелой цепи и вариабельные области легкой цепи:
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 1, CDR2 тяжелой цепи по SEQ ID NO: 8 и CDR3 тяжелой цепи по SEQ ID NO: 16, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 88, CDR2 легкой цепи по SEQ ID NO: 103 и CDR3 легкой цепи по SEQ ID NO: 120;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 121;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи SEQ ID NO: 2, CDR2 тяжелой цепи SEQ ID NO: 9 и CDR3 тяжелой цепи SEQ ID NO: 18, и вариабельную область легкой цепи, включающую CDR1 легкой цепи SEQ ID NO: 90, CDR2 легкой цепи SEQ ID NO: 105 и CDR3 легкой цепи SEQ ID NO: 122;
по вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи SEQ ID NO: 3, CDR2 тяжелой цепи SEQ ID NO: 10 и CDR3 тяжелой цепи SEQ ID NO: 19, и вариабельную область легкой цепи, включающую CDR1 легкой цепи SEQ ID NO: 91, CDR2 легкой цепи по SEQ ID NO: 106 и CDR3 легкой цепи по SEQ ID NO: 123;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 4, CDR2 тяжелой цепи по SEQ ID NO: 11 и CDR3 тяжелой цепи по SEQ ID NO: 20, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 107 и CDR3 легкой цепи по SEQ ID NO: 124;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 5, CDR2 тяжелой цепи по SEQ ID NO: 12 и CDR3 тяжелой цепи по SEQ ID NO: 21, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 92, CDR2 легкой цепи по SEQ ID NO: 108 и CDR3 легкой цепи по SEQ ID NO: 122;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 6, CDR2 тяжелой цепи по SEQ ID NO: 13 и CDR3 тяжелой цепи по SEQ ID NO: 22, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 93, CDR2 легкой цепи по SEQ ID NO: 109 и CDR3 легкой цепи по SEQ ID NO: 125;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 23, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 94, CDR2 легкой цепи по SEQ ID NO: 110 и CDR3 легкой цепи по SEQ ID NO: 126;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 7, CDR2 тяжелой цепи по SEQ ID NO: 14 и CDR3 тяжелой цепи по SEQ ID NO: 24, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 95, CDR2 легкой цепи по SEQ ID NO: 111 и CDR3 легкой цепи по SEQ ID NO: 127; или
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 15 и CDR3 тяжелой цепи по SEQ ID NO: 25, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 96, CDR2 легкой цепи по SEQ ID NO: 112 и CDR3 легкой цепи по SEQ ID NO: 128.
В соответствии с одним воплощением настоящего изобретения, антитела далее подвергаются скринингу по процедуре оптимизации, причем антитело либо его антигенсвязывающий фрагмент по изобретению может содержать следующие вариабельные области тяжелой цепи и вариабельные области легкой цепи:
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 108 и CDR3 легкой цепи по SEQ ID NO: 129;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 130;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 113 и CDR3 легкой цепи по SEQ ID NO: 131;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 97, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 132;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 133;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 97, CDR2 легкой цепи по SEQ ID NO: 114 и CDR3 легкой цепи по SEQ ID NO: 134;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 92, CDR2 легкой цепи по SEQ ID NO: 115 и CDR3 легкой цепи по SEQ ID NO: 135;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 98, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 130;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 116 и CDR3 легкой цепи по SEQ ID NO: 121;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 108 и CDR3 легкой цепи по SEQ ID NO: 136;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 99, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 137;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 117 и CDR3 легкой цепи по SEQ ID NO: 138;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 118 и CDR3 легкой цепи по SEQ ID NO: 133;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 119 и CDR3 легкой цепи по SEQ ID NO: 139;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 100, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 140;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 108 и CDR3 легкой цепи по SEQ ID NO: 141;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 139;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 142;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 143;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 101, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 141; или
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 102, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 144.
В частности, антитело либо его антигенсвязывающий фрагмент по изобретению может содержать следующие вариабельные области тяжелой цепи и вариабельные области легкой цепи:
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 121;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 130;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 104 и CDR3 легкой цепи по SEQ ID NO: 133;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 108 и CDR3 легкой цепи по SEQ ID NO: 136;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 119 и CDR3 легкой цепи по SEQ ID NO: 139;
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 139; или
вариабельную область тяжелой цепи, включающую CDR1 тяжелой цепи по SEQ ID NO: 2, CDR2 тяжелой цепи по SEQ ID NO: 9 и CDR3 тяжелой цепи по SEQ ID NO: 17, и вариабельную область легкой цепи, включающую CDR1 легкой цепи по SEQ ID NO: 89, CDR2 легкой цепи по SEQ ID NO: 105 и CDR3 легкой цепи по SEQ ID NO: 143.
Термин “каркасный участок” (FR) относится к остаткам вариабельного домена, отличным от остатков CDR. Каждый вариабельный домен обычно содержит четыре FR, обозначаемых как FR1, FR2, FR3 и FR4.
В соответствии с одним воплощением настоящего изобретения, антитела либо их антигенсвязывающие фрагменты могут включать в себя:
FR1 вариабельной области тяжелой цепи, выбранный из группы, состоящей из SEQ ID NOs: 26-34;
FR2 вариабельной области тяжелой цепи, выбранный из группы, состоящей из SEQ ID NOs: 35-41;
FR3 вариабельной области тяжелой цепи, выбранный из группы, состоящей из SEQ ID NOs: 42-49; или
FR4 вариабельной области тяжелой цепи, выбранный из группы, состоящей из SEQ ID NOs: 50-54.
Кроме того, антитело либо его антигенсвязывающий фрагмент может включать в себя:
FR1 вариабельной области легкой цепи, выбранный из группы, состоящей из SEQ ID NOs: 145-163;
FR2 вариабельной области легкой цепи, выбранный из группы, состоящей из SEQ ID NOs: 164-184;
FR3 вариабельной области легкой цепи, выбранный из группы, состоящей из SEQ ID NOs: 185-210; или
FR4 вариабельной области легкой цепи, выбранный из группы, состоящей из SEQ ID NOs: 211-216.
Фрагмент “Fv” представляет собой фрагмент антитела, содержащий полные сайты распознавания и связывания антител. Такая область включает димер, к примеру, scFv, который состоит из одного вариабельного домена тяжелой цепи и одного вариабельного домена легкой цепи, весьма крепко связанных ковалентно друг с другом.
“Fab”-фрагмент содержит вариабельный и константный домены легкой цепи и вариабельный и первый константный домен (CH1) тяжелой цепи. F(ab′)2-фрагмент антител обычно включает пару Fab-фрагментов, ковалентно связанных через шарнирный цистеин, расположенный между ними вблизи карбоксильного конца.
“Одноцепочечный Fv” или “scFv”-фрагмент антитела включает домены VH и VL антител, причем эти домены присутствуют в виде одной полипептидной цепи. Полипептид Fv может дополнительно содержать полипептидный линкер между доменом VH и доменом VL для того, чтобы scFv образовывал структуру, необходимую для связывания антигена.
Антитела к PD-L1 бывают моновалентными или бивалентными и включают в себя короткие или двойные цепи. Функционально, сродство связывания антител к PD-L1 составляет от 10-5 M до 10-12 M. К примеру, сродство связывания антител к PD-L1 составляет от 10-6 M до 10-12 M, от 10-7 M до 10-12 M, от 10-8 M до 10-12 M, от 10-9 M до 10-12 M, от 10-5 M до 10-11 M, от 10-6 M до 10-11 M, от 10-7 M до 10-11 M, от 10-8 M до 10-11 M, от 10-9 M до 10-11 M, от 10-10 M до 10-11 M, от 10-5 M до 10-10 M, от 10-6 M до 10-10 M, от 10-7 M до 10-10 M, от 10-8 M до 10-10 M, от 10-9 M to 10-10 M, от 10-5 M to 10-9 M, от 10-6 M до 10-9 M, от 10-7 M to 10-9 M, 10-8 M до 10-9 M, 10-5 M до 10-8 M, 10-6 M до 10-8 M, от 10-7 M до 10-8 M, от 10-5 M до 10-7 M, от 10-6 M до 10-7 M или от 10-5 M до 10-6 M.
Антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты могут содержать вариабельную область тяжелой цепи, включающую последовательность, которая на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 57-87. Антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты могут содержать вариабельную область тяжелой цепи, выбранную из группы, состоящей из SEQ ID NOs: 57-87. В одном воплощении настоящего изобретения антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты могут содержать вариабельную область тяжелой цепи по SEQ ID NO: 58, 68, 71, 76, 80, 83 или 85.
Кроме того, антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты могут содержать вариабельную область легкой цепи, включающую последовательность, которая на 90% или больше идентична последовательности, выбранной из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 217-247. Антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты могут содержать вариабельную область легкой цепи, выбранную из группы, состоящей из последовательностей, приведенных в SEQ ID NOs: 217-247. В одном воплощении настоящего изобретения антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты могут содержать вариабельную область легкой цепи по SEQ ID NO: 218, 228, 231, 236, 240, 243 или 245.
В одном конкретном воплощении настоящего изобретения антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты могут включать в себя:
вариабельную область тяжелой цепи по SEQ ID NO: 58 и вариабельную область легкой цепи по SEQ ID NO: 218;
вариабельную область тяжелой цепи по SEQ ID NO: 68 и вариабельную область легкой цепи по SEQ ID NO: 228;
вариабельную область тяжелой цепи по SEQ ID NO: 71 и вариабельную область легкой цепи по SEQ ID NO: 231;
вариабельную область тяжелой цепи по SEQ ID NO: 76 и вариабельную область легкой цепи по SEQ ID NO: 236;
вариабельную область тяжелой цепи по SEQ ID NO: 80 и вариабельную область легкой цепи по SEQ ID NO: 240;
вариабельную область тяжелой цепи по SEQ ID NO: 83 и вариабельную область легкой цепи по SEQ ID NO: 243; или
вариабельную область тяжелой цепи по SEQ ID NO: 85 и вариабельную область легкой цепи по SEQ ID NO: 245.
“Фаговый дисплей” представляет собой метод представления мутантных полипептидов в виде слитых белков с по меньшей мере частью белка оболочки, к примеру, на поверхности частиц фага, к примеру, филаментного фага. Полезность фагового дисплея состоит в быстрой и эффективной классификации тех последовательностей, которые связываются с целевыми антигенами с высоким сродством, в больших библиотеках рандомизованных мутантов белка. Выставление пептидов и библиотек белков на фагах применяется для скрининга миллионов полипептидов с тем, чтобы идентифицировать полипептиды с определенными свойствами связывания.
Технология фагового дисплея оказалась мощным инструментом для создания и скрининга новых белков, связывающихся с определенными лигандами (например, антигенами). По технологии фагового дисплея можно создавать большие библиотеки мутантов белка и быстро классифицировать последовательности, связывающиеся с целевыми антигенами с высоким сродством. Нуклеиновую кислоту, кодирующую мутантный полипептид, сливают с последовательностью нуклеиновой кислоты, кодирующей белки оболочки вируса, например, белки гена III или белки гена VIII. Разработана монофазная система фагового дисплея, в которой последовательность нуклеиновой кислоты, кодирующей белок или полипептид, сливают с последовательностью нуклеиновой кислоты, кодирующей часть белка гена III. В монофазной системе дисплея слитый ген экспрессируется на низком уровне, также экспрессируется белок гена III дикого типа, при этом сохраняется инфекционность частиц.
При разработке библиотек антител типа фагового дисплея важно продемонстрировать экспрессию пептидов на поверхности филаментного фага и экспрессию функциональных фрагментов антител в периферической цитоплазме E. coli. Библиотеки антител либо антигенсвязывающих полипептидов получают рядом методов, к примеру, путем модификации одного гена при вставке случайной последовательности ДНК или клонирования последовательности родственного гена. Библиотеки можно подвергать скринингу на экспрессию антител или антигенсвязывающих белков с требуемыми характеристиками.
Технология фагового дисплея имеет ряд преимуществ перед стандартными гибридомными и рекомбинантными методами получения антител с нужными характеристиками. Этот метод обеспечивает получение больших библиотек антител с разнообразными последовательностями за короткое время без использования животных. Для получения гибридом и получения гуманизованных антител может потребоваться время в несколько месяцев. Кроме того, поскольку иммунитет не требуется, то фаговые библиотеки антител могут вырабатывать антитела против антигенов, которые являются токсичными или обладают слабой антигенностью. Фаговые библиотеки антител также можно использовать для получения и идентификации новых терапевтических антител.
Для получения человеческих антител от неиммунизованных людей этим методом можно использовать гаметные последовательности или Ig-репертуары наивных В-клеток, иммунизированных с помощью библиотек фагового дисплея. Для получения библиотек нативных или неиммуногенных антигенсвязывающих полипептидов можно использовать различные лимфатические ткани.
Для выделения новых терапевтических антител из библиотек фагового дисплея важны методы идентификации и выделения антител с высоким сродством. Выделение антител с высоким сродством из библиотек может зависеть от размера библиотеки, эффективности продукции в бактериальных клетках и разнообразия библиотек. Размер библиотек уменьшается при неэффективной укладке антител или антигенсвязывающих белков и неэффективной продукции из-за присутствия стоп-кодона. Экспрессия в бактериальных клетках может ингибироваться при неправильной укладке антител или антигенсвязывающих доменов. Экспрессия может быть улучшена путем попеременного мутирования остатков на поверхности границ вариабельных/константных областей или выбранных остатков CDR. Последовательность каркасных участков является тем элементом, который обеспечивает надлежащую укладку при получении фаговых библиотек антител в бактериальных клетках.
При выделении антител с высоким сродством важно создавать разнообразные библиотеки антител или антигенсвязывающих белков. Участки CDR3, как оказалось, зачастую участвуют в связывании антигена. Поскольку участки CDR3 в тяжелой цепи значительно различаются по размерам, последовательности и структурно-пространственной морфологии, то с их помощью можно получать разнообразные библиотеки.
Кроме того, можно создавать разнообразие путем рандомизации участков CDR у вариабельных тяжелых и легких цепей, используя все 20 аминокислот в каждом положении. Использование всех 20 аминокислот ведет к получению последовательностей антител с повышенным разнообразием и повышенной вероятностью выявления новых антител.
Антитела или фрагменты антител по настоящему изобретению могут включать в себя описанные здесь последовательности антител против PD-L1 по настоящему изобретению, а также их биологические эквиваленты, если только эти антитела или фрагменты антител могут специфически распознавать PD-L1. Например, в аминокислотной последовательности антитела могут производиться дополнительные вариации с тем, чтобы еще больше улучшить сродство связывания и/или другие биологические свойства антитела. Такие вариации включают, к примеру, делеции, вставки и/или замены остатков в аминокислотной последовательности антитела. Такие вариации аминокислот производятся на основании относительного сходства (идентичности) заместителей в боковой цепи аминокислот типа гидрофобности, гидрофильности, заряда или размера. Анализ размеров, формы и типа заместителей в боковой цепи аминокислот показывает, что аргинин, лизин и гистидин являются положительно заряженными остатками, аланин, глицин и серин имеют близкие размеры, а фенилаланин, триптофан и тирозин имеют близкие формы. Так, исходя из этих соображений, аргинин, лизин и гистидин; аланин, глицин и серин; фенилаланин, триптофан и тирозин считаются биологически функционально эквивалентными.
При рассмотрении вариантов, обладающих биологически эквивалентной активностью, антитела либо кодирующие их нуклеиновые кислоты по настоящему изобретению также могут включать в себя последовательности, проявляющие существенную идентичность с последовательностью, приведенной в соответствующем SEQ ID NO. Термин “последовательность, проявляющая существенную идентичность” означает, что последовательность проявляет идентичность по меньшей мере на 90%, наиболее предпочтительно по меньшей мере на 95%, 96% и более, 97% и более, 98% и более или 99% и более при совмещении последовательности по настоящему изобретению таким образом, чтобы она как можно больше соответствовала какой-либо другой последовательности, и анализе совмещенных последовательностей с помощью алгоритма, который обычно применяется в данной области. Методы совмещения для сравнения последовательностей хорошо известны в данной области. Базовый инструмент поиска локального совмещения NCBI (BLAST) доступен из NBCI и может применяться вместе с такими программами анализа последовательностей, как blastp, blasm, blastx, tblastn и tblastx в Интернете. BLAST доступен по адресу www.ncbi.nlm.nih.gov/BLAST/. Метод сравнения последовательностей по идентичности с помощью этой программы находится по адресу www.ncbi.nlm.nih.gov/BLAST/blast_help.html.
Исходя из этого, антитела либо их антигенсвязывающие фрагменты по настоящему изобретению могут проявлять идентичность (гомологичность) последовательности в 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% и больше. Такая идентичность определяется путем сравнения и/или совмещения последовательностей методами, известными в данной области. Например, степень идентичности последовательности нуклеиновой кислоты или белка по настоящему изобретению можно определить с помощью алгоритма сравнения последовательностей (т.е. BLAST или BLAST 2.0), совмещения вручную или визуального осмотра.
В другом аспекте настоящее изобретение касается нуклеиновых кислот, кодирующих антитела либо их антигенсвязывающие фрагменты.
Антитело либо его антигенсвязывающий фрагмент может быть получены рекомбинантным способом путем выделения нуклеиновой кислоты, кодирующей антитело либо его антигенсвязывающий фрагмент по настоящему изобретению. Нуклеиновую кислоту выделяют и вставляют в реплицирующийся вектор для дальнейшего клонирования (амплификации ДНК) или дальнейшей экспрессии. Исходя из этого, в другом аспекте настоящее изобретение касается вектора, содержащего нуклеиновую кислоту.
Термин “нуклеиновая кислота” служит для обозначения молекул ДНК (гДНК и кДНК) и РНК, а нуклеотиды, которые являются основными составляющими единицами нуклеиновой кислоты, включают как нуклеотиды природного происхождения, так и их аналоги, у которых модифицированы молекулы сахаридов или оснований. Последовательность нуклеиновой кислоты, кодирующей вариабельные области тяжелой и легкой цепи по настоящему изобретению, можно варьировать. Такие вариации включают добавление, делеции либо неконсервативные или консервативные замены нуклеотидов.
ДНК, кодирующая антитело, может быть легко выделена или синтезирована по стандартной методике (например, с помощью олигонуклеотидного зонда, специфически связывающегося с ДНК, кодирующей тяжелые и легкие цепи антитела). Доступны различные векторы. Компоненты векторов обычно включают, без ограничения, один или несколько из следующих компонентов: сигнальные последовательности, точки начала репликации, один или несколько маркерных генов, элементы энхансеров, промоторы и последовательности терминации транскрипции.
В настоящем изобретении термин “вектор” означает средство для экспрессии целевых генов в клетках хозяина и включает: плазмидные векторы; космидные векторы; и вирусные векторы, как-то бактериофаговые векторы, аденовирусные векторы, ретровирусные векторы и аденоассоциированные вирусные векторы. Нуклеиновая кислота, кодирующая антитело в векторе, оперативно связана с промотором.
Термин “оперативно связанный” означает функциональную связь между последовательностью, регулирующей экспрессию нуклеиновой кислоты (например, промотором, сигнальной последовательностью или набором сайтов связывания регуляторов транскрипции), и другой последовательностью нуклеиновой кислоты, которая регулируется при транскрипции и/или трансляции последовательности нуклеиновой кислоты.
При использовании в качестве хозяина прокариотических клеток вектор обычно содержит сильный промотор, способный осуществлять транскрипцию (как-то промотор tac, промотор lac, промотор lacUV5, промотор lpp, промотор pLλ, промотор pRλ, промотор rac5, промотор amp, промотор recA, промотор SP6, промотор trp или промотор T7), сайт связывания рибосомы для инициации трансляции и последовательность терминации транскрипции/трансляции. Кроме того, к примеру, при использовании в качестве хозяина эукариотических клеток вектор содержит промотор (например, промотор металлотионеина, промотор β-актина, промотор гемоглобина человека или промотор мышечного креатина человека), полученный из генома клеток млекопитающих, промотор, происходящий из вируса животных, как-то поздний промотор аденовируса, промотор 7.5K вируса коровьей оспы, промотор SV40, промотор цитомегаловируса (CMV), tk-промотор HSV, промотор вируса опухолей молочной железы мыши (MMTV), промотор LTR ВИЧ, промотор вируса Молони, промотор вируса Эпштейна-Барра (EBV) и промотор вируса саркомы Рауса (RSV), а также обычно содержит последовательность полиаденилирования в качестве последовательности терминации транскрипции.
Необязательно вектор может быть слит с другой последовательностью для облегчения очистки экспрессируемых из него антител. Последовательности для слияния включают, к примеру, глутатион-S-трансферазу (Pharmacia, USA), мальтозосвязывающий белок (NEB, USA), FLAG (IBI, USA), 6xHis (гексагистидин; Quiagen, USA) и др.
Вектор содержит гены устойчивости к антибиотикам, широко используемые в данной области в качестве отборочных маркеров, примеры которых включают гены устойчивости к ампициллину, гентамицину, карбенициллину, хлорамфениколу, стрептомицину, канамицину, генетицину, неомицину и тетрациклину.
В другом аспекте настоящее изобретение касается клетки, трансформированной вышеупомянутым вектором. Клетка, используемая для получения антитела по настоящему изобретению, может представлять собой прокариотическую, дрожжевую клетку или высшую эукариотическую клетку, без ограничения.
Можно использовать штаммы из рода Bacillus типа Escherichia coli, Bacillus subtilis и Bacillus tuligensis, Streptomyces, Pseudomonas (к примеру, Pseudomonas putida) и такие прокариотические клетки хозяина, как Proteus mirabilis и Staphylococcus (к примеру, Staphylococcus carnosus).
Наибольший интерес представляют клетки животных, а примеры полезных линий клеток хозяина включают, без ограничения, COS-7, BHK, CHO, CHOK1, DXB-11, DG-44, CHO/DHFR, CV1, COS-7, HEK293, BHK, TM4, VERO, HELA, MDCK, BRL 3A, W138, Hep G2, SK-Hep, MMT, TRI, MRC 5, FS4, 3T3, RIN, A549, PC12, K562, PER.C6, SP2/0, NS-0, U20S или HT1080.
В другом аспекте настоящее изобретение касается способа получения антител либо их антигенсвязывающих фрагментов, включающего: (a) культивирование клеток; и (b) выделение антител либо их антигенсвязывающих фрагментов из культуры клеток.
Клетки можно культивировать в различных средах. В качестве культуральной среды можно использовать любые коммерчески доступные среды без ограничения. Все другие необходимые добавки, хорошо известные специалистам в данной области, могут быть включены в соответствующих концентрациях. Условия культивирования типа температуры и pH уже использовались с выбранными для экспрессии клетками хозяина и должны быть известны специалистам в данной области.
Выделение антител либо их антигенсвязывающих фрагментов может проводиться, к примеру, путем центрифугирования или ультрафильтрации для удаления примесей и очистки полученного препарата, к примеру, методом аффинной хроматографии. Можно использовать и другие методы очистки типа анионообменной или катионообменной хроматографии, хроматографии гидрофобных взаимодействий, хроматографии на гидроксиапатите и др.
В другом аспекте настоящее изобретение касается получение композиции для профилактики или лечения рака, содержащей антитело в качестве активного ингредиента.
Настоящим изобретением предусмотрены, к примеру, композиции для профилактики или лечения рака либо инфекционных заболеваний, содержащие: (a) фармацевтически эффективное количество антитела к PD-L1 или его антигенсвязывающего фрагмента по изобретению; и (b) фармацевтически приемлемый носитель. Настоящим изобретением также предусмотрен способ профилактики или лечения рака или инфекционных заболеваний, включающий введение антитела к PD-L1 либо его антигенсвязывающего фрагмента по настоящему изобретению в эффективном количестве, необходимом для пациента.
Поскольку в качестве активного ингредиента в композиции применяется антитело против PD-L1 либо его антигенсвязывающий фрагмент по настоящему изобретению, описанные выше, то их повторное описание опущено.
Связывание PD-L1 с PD-1 вызывает отрицательную регуляцию антиген-специфичных реакций Т-клеток, важных для толерантности и предотвращения аутоиммунитета и иммунопатологии. Однако чрезмерное взаимодействие PD-L1/PD-1, которое может возникать при хронической стимуляции антигенами, может вызвать ингибирование антиген-специфичных реакций Т-клеток и потерю Т-клеток, что характерно для истощения Т-клеток. Истощение Т-клеток – это состояние дисфункции Т-клеток, которое может возникать при хронических инфекциях и раковых заболеваниях. Истощение Т-клеток определяется как ухудшение эффекторной функции, непрерывная экспрессия ингибиторных рецепторов или состояние транскрипции, отличающееся от функциональных эффекторов или Т-клеток памяти. Истощение препятствует развитию инфекций и опухолей.
Как показано в нижеследующих примерах, антитела либо их антигенсвязывающие фрагменты по изобретению связываются с PD-L1 с высоким сродством и ингибируют образование комплекса PD-1 и PD-L1, поэтому они применимы для лечения рака, вызывая истощение Т-клеток в обход противоопухолевой активности Т-клеток.
В некоторых случаях в комбинации с ними можно использовать другие противораковые терапевтические средства, отличные от вышеупомянутых антител, для эффективного воздействия на опухолевые клетки, гиперэкспрессирующие PD-L1, чтобы усилить противоопухолевую активность Т-клеток и тем самым улучшить иммунный ответ, нацеленный на опухолевые клетки. Вышеупомянутые антитела можно использовать в комбинации с другими противоопухолевыми или иммуногенными средствами (к примеру, ослабленными раковыми клетками, опухолевыми антигенами, включая рекомбинантные белки, пептиды и молекулы углеводов, антигенпрезентирующими клетками типа дендритных клеток, обработанных опухолевыми антигенами или нуклеиновыми кислотами, клетками, трансфецированными иммуностимулирующим цитокином (например, IL-2, IFNα2, GM-CSF), и генами, кодирующими иммуностимулирующие цитокины (включая, без ограничения, GM-CSF); стандартной противораковой терапией (например, химиотерапией, лучевой терапией или хирургией) или другими антителами (включая, без ограничения, VEGF, EGFR, Her2/neu, рецепторы VEGF, рецепторы других факторов роста, CD20, CD40, CTLA-4, OX-40, 4-IBB и ICOS).
Антитела против PD-L1 могут вызывать апоптоз (клеточную смерть). Апоптоз индуцируется по прямым или косвенным механизмам. Например, связывание антител против PD-L1 с PD-L1 может вызывать комплемент-зависимую цитотоксичность (CDC). В некоторых случаях антитела против PD-L1 связываются с PD-L1 и вызывают мобилизацию вторичных типов клеток, уничтожающих целевые клетки, экспрессирующие PD-L1. Репрезентативные механизмы, по которым антитела против PD-L1 опосредуют апоптоз путем мобилизации вторичных типов клеток, включают, без ограничения, антителозависимую цитотоксичность (ADCC) и антителозависимую клеточную цитотоксичность (ADCP). Целевые типы клеток, экспрессирующие PD-L1, включают опухоли и Т-клетки типа активированных Т-клеток.
Кроме того, антитела либо их фрагменты по настоящему изобретению могут применяться для профилактики или лечения инфекций и инфекционных заболеваний.
В настоящем изобретении термин “профилактика” означает любое действие, которое подавляет раковые или инфекционные заболевания либо замедляет их прогрессирование путем введения композиции, а термин “лечение” в настоящем изобретении означает торможение развития рака либо облегчение или устранение рака или же подавление, облегчение или устранение инфекционных заболеваний.
Рак, то есть заболевание, при котором применяются композиции, обычно включает рак, который реагирует на иммунотерапию, и рак, который до сих пор не был вовлечен в иммунотерапию. Неограничительные примеры предпочтительных раковых заболеваний, подлежащих лечению, включают, без ограничения, меланому (например, метастатическую злокачественную меланому), рак почек (например, светлоклеточную карциному), рак простаты (например, гормонорефрактерную аденокарциному простаты), аденокарциному поджелудочной железы, рак молочной железы, рак толстой кишки, рак легких (например, немелкоклеточный рак легких), рак пищевода, плоскоклеточную карциному головы и шеи, рак печени, рак яичников, рак шейки матки, рак щитовидной железы, глиобластому, глиому, лейкоз, лимфому и другие неопластические карциномы. Кроме того, настоящее изобретение охватывает рефрактерные или рецидивирующие раковые опухоли, рост которых можно ингибировать с помощью антител по изобретению.
Антитела или фрагменты антител можно использовать по отдельности или же в комбинации с вакциной для стимуляции иммунного ответа на патогены, токсины и аутоантигены. Антитела либо их антигенсвязывающие фрагменты можно использовать для стимуляции иммунных реакций на вирусы, заражающие людей, включая, без ограничения, вирус иммунодефицита человека, вирусы гепатита A, B и C, вирус Эпштейна-Барра, цитомегаловирус человека, вирусы папилломы и герпеса человека. Антитела либо их антигенсвязывающие фрагменты можно использовать для стимуляции иммунных реакций на заражение бактериальными или грибковыми паразитами и другими патогенами.
Фармацевтически приемлемые носители, которые содержатся в композициях по настоящему изобретению, включают, без ограничения, лактозу, декстрозу, сахарозу, сорбит, маннит, крахмал, гуммиарабик, фосфат кальция, альгинат, желатин, силикат кальция, микрокристаллическую целлюлозу, поливинилпирролидон, воду, сиропы, метилцеллюлозу, метилгидроксибензоат, пропилгидроксибензоат, тальк, стеарат магния, минеральное масло и др., которые обычно применяются для приготовления лекарств. Наряду с вышеуказанными компонентами, композиции по настоящему изобретению также могут содержать смазывающие, смачивающие вещества, подсластители, ароматизаторы, эмульгаторы, суспендирующие вещества, консерванты и т.п.
Фармацевтические композиции по настоящему изобретению можно вводить перорально или парентерально. В случае парентерального введения фармацевтические композиции можно вводить внутривенно, подкожно, внутримышечно, внутрибрюшинно, эндотелиально, местно, интраназально, внутрилегочно, ректально и т.п.
При пероральном введении белок или пептид может перевариваться. По этой причине пероральные композиции следует составлять так, чтобы покрывать активный ингредиент или защищать белок или пептид от переваривания в желудке. Кроме того, фармацевтические композиции можно вводить при помощи любых устройств, способных доставлять активный ингредиент в целевые клетки.
Подходящая дозировка композиций по настоящему изобретению может варьироваться в зависимости от таких факторов, как способ составления, способ введения, возраст, вес тела, пол, заболевание и питание пациента, время введения, способ введения, скорость выведения и восприимчивость. Опытный врач может легко определить и назначить дозировку, эффективную для данного лечения или профилактики. Например, суточная дозировка фармацевтической композиции настоящего изобретения может составлять от 0,0001 до 100 мг/кг. В настоящем изобретении термин “фармацевтически эффективное количество” означает количество, достаточное для профилактики или лечения рака.
Фармацевтические композиции настоящего изобретения могут быть приготовлены в виде стандартных дозовых форм или заключены во флаконы с множественными дозами в виде лекарственных форм с фармацевтически приемлемыми носителями и/или эксципиентами в соответствии с такими способами, которые легко выполнимы рядовыми специалистами в той области техники, к которой относится настоящее изобретение. Композиции могут быть в виде растворов, суспензий либо эмульсий в масле или водной среде или же в виде наполнителей, порошков, свечей, порошков, гранул, таблеток или капсул и также могут содержать диспергирующие или стабилизирующие средства.
Композиции по настоящему изобретению могут вводиться в виде отдельного терапевтического средства или в комбинации с другими терапевтическими средствами и могут вводиться последовательно или одновременно со стандартными терапевтическими средствами.
В другом аспекте настоящее изобретение касается композиций для диагностики рака, содержащих антитела к PD-L1 либо их антигенсвязывающие фрагменты по настоящему изобретению. Кроме того, настоящее изобретение касается способа диагностики рака путем обработки антителами к PD-L1 либо их антигенсвязывающими фрагментами по настоящему изобретению.
Рак можно диагностировать путем измерения уровня экспрессии PD-L1 в образцах с помощью антител к PD-L1 по настоящему изобретению. Уровень экспрессии может быть измерен стандартным методом иммуноанализа, включая, без ограничения, методы радиоиммуноанализа, радиоиммунопреципитации, иммунопреципитации, иммуногистохимического окрашивания, иммуноферментного анализа (ELISA), ELISA с захватом, анализа ингибирования или конкуренции, сэндвич-анализа, проточной цитометрии, иммунофлуоресцентного окрашивания и иммуноаффинной очистки с помощью антител к PD-L1.
Рак можно диагностировать путем анализа интенсивности конечного сигнала в процессе иммуноанализа. А именно, рак диагностируется тогда, когда в биологическом образце сильно экспрессируется белок маркера по настоящему изобретению, при этом сигнал от биологического образца будет более сильным, чем у нормального биологического образца (к примеру, нормальной ткани желудка, крови, плазмы или сыворотки).
В другом аспекте настоящее изобретение касается наборов для диагностики рака, содержащих композиции для диагностики рака. Наборы по настоящему изобретению содержат антитела к PD-L1 по настоящему изобретению, а рак диагностируется путем анализа сигнала, возникающего при реакции между образцом и антителом. Сигналы могут включать, без ограничения, конъюгированные с антителами ферменты типа щелочной фосфатазы, β-галактозидазы, пероксидазы хрена, люциферазы или цитохрома P450. В том случае, когда в качестве фермента используется щелочная фосфатаза, в качестве субстрата для фермента используется хромогенный субстрат реакции, как-то бромхлориндолфосфат (BCIP), нитросиний тетразолий (NBT), нафтол-AS-B1-фосфат или ECF (для усиления хемифлуоресценции), а при использовании пероксидазы хрена используется такой субстрат, как хлорнафтол, аминоэтилкарбазол, диаминобензидин, D-люциферин, люцигенин (бис-N-метилакридиния нитрат), бензиловый эфир резоруфина, люминол, реагент Amplex Red (10-ацетил-3,7-дигидроксифеноксазин), HYR (п-фенилендиамин-HCl и пирокатехол), TMB (тетраметилбензидин), ABTS (2,2′-азин-ди[3-этилбензтиазолинсульфонат]), о-фенилендиамин (OPD) и нафтол/пиронин, глюкозооксидаза, t-NBT (нитросиний тетразолий) или m-PMS (феназин метосульфат), но настоящее изобретение этим не ограничивается.
Кроме того, набор по настоящему изобретению также может включать метку для генерирования детектируемого сигнала, а метка может означать химическое вещество (например, биотин), фермент (щелочная фосфатаза, β-галактозидаза, пероксидаза хрена или цитохром P450), радиоактивное вещество (типа 14C, 125I, 32P и 35S), флуоресцентное вещество (типа флуоресцеина), люминесцентное вещество, хемилюминесцентное вещество и FRET (флуоресцентно-резонансный перенос энергии), без ограничения.
Измерение активности фермента, используемого для диагностики рака или измерения сигнала, может проводиться различными способами, известными в данной области. Таким образом, можно качественно или количественно анализировать экспрессию PD-L1.
Примеры
Далее настоящее изобретение будет описано более подробно с привлечением примеров. Однако специалистам в данной области должно быть ясно, что эти примеры приводятся только для иллюстрации настоящего изобретения и не должны рассматриваться как ограничивающие объем настоящего изобретения.
Пример 1. Экспрессия и очистка антигена PD-L1
1. Получение векторов для экспрессии белка PD-L1
Для клонирования PD-L1 проводили амплификацию методом полимеразной цепной реакции (ПЦР) с помощью праймеров для PD-L1, содержащих сайты для рестрикционного фермента SfiI на 5′- и 3′-концах (таблица 1) с тем, чтобы получить только внеклеточный домен, используя библиотеки кДНК в клетках Jurkat (Stratagene, USA). Амплифицированный продукт ПЦР получали путем слияния Fc человека (SEQ ID NO: 248) и Fc мыши (SEQ ID NO: 249) с карбоксильным концом с помощью вектора N293F (фиг. 1).
Таблица 1. Праймеры для векторов, экспрессирующих PD-L1
2. Экспрессия и очистка антигена PD-L1
Для экспрессирования антигена в клетках животных трансфецировали клетки HEK293F плазмидной ДНК. Готовили полиплексный реакционный раствор для трансфекции путем смешивания 25 мкг плазмидной ДНК с 3 мл экспрессионной среды Freestyle 293, а затем в полученную смесь добавляли 2 мг/мл PET (полиэтиленимин, PolyplusA-transfection, USA) и опять перемешивали. Полиплексный реакционный раствор инкубировали при комнатной температуре в течение 15 мин, а затем культивировали в 40 мл культуральной среды (1×106 клеток/мл) в течение 24 часов при 37°C и 8% CO2 при 120 об/мин. Через 24 часа после трансфекции добавляли Soytone (BD, USA) в качестве добавки до конечной концентрации 10 г/л. Антитела получали в системе краткосрочной экспрессии в клетках HEK293F в течение 7 дней. Для выделения антигена из культуральной среды проводили аффинную хроматографию. Получали супернатант центрифугированием при 5000 об/мин в течение 10 мин для удаления клеток и обломков клеток из культуральной среды, извлеченной на 7-й день. Супернатант подвергали реакции с промытой DPBS агарозной смолой с рекомбинантным белком A при 4°C в течение 16 часов.
При использовании агарозной смолы с рекомбинантным белком A для проведения первичной очистки белок элюировали 0,1 М глицином и нейтрализировали 500 мкл 1М трис-HCl. После первичной очистки белок подвергали вторичной очистке методом гель-фильтрационной хроматографии на Superdex 200 (1,5 см ×100 см).
Чистоту очищенного белка определяли методом SDS-PAGE и эксклюзионной хроматографии [TSK-GEL G-3000 SWXL для эксклюзионной хроматографии (SEC) (Tosoh)].
В результате было установлено, что очищенный белок PD1 имеет чистоту в 95% или больше, как видно из фиг. 2А–2D.
Пример 2. Скрининг человеческих антител против PD-L1
1. Подготовка антигенов
В качестве белковых антигенов PD-L1-hFc и PD-L1-mFc, полученные в примере 1, и PD-L1-his (кат. № 10377-H08H), приобретенный у Sino Biological Inc., фиксировали в дозе 50 мкг на иммуносорбционных пробирках, а затем блокировали.
2. Биопэннинг (просеивание)
Получали фаговую библиотеку человеческих антител путем инфицирования бактерий библиотекой scFv человека с разнообразием в 2,7×1010, а затем культивирования при 30°C в течение 16 ч. Культуральную среду после культивирования центрифугировали, а супернатант концентрировали с помощью PEG и затем растворяли в буфере PBS для получения библиотеки человеческих антител. Фаговую библиотеку человеческих антител вносили в иммунологическую пробирку и инкубировали при комнатной температуре в течение 2 часов. После отмывки 1 раз PBS/T и 1 раз PBS элюировались только scFv-фаги, специфически связавшиеся с антигеном. Элюированными фагами опять инфицировали E. coli и проводили амплификацию (процесс пэннинга), получая пул положительных фагов. Проводили второй и третий раунд пэннинга, используя фаги, амплифицированные в первом раунде пэннинга, таким же образом, как описано выше, за исключением того, что увеличилось только количество стадий промывки PBST.
В результате, как видно из таблицы 2, оказалось, что количество фагов, связавшихся с антигеном (на выходе) при третьем раунде пэннинга, немного повысилось по сравнению с количеством фагов на входе.
Таблица 2. Сравнение титра антител в зависимости от количества раундов пэннинга
3. Полифаговый метод ELISA
Исходные клетки, замороженные после каждого из трех раундов пэннинга, вносили в среду, содержащую 5 мл 2×YTCM, 2% глюкозы и 5 мМ MgCl2, до значения OD600 = 0,1, а затем культивировали при 37°C в течение 2-3 часов (OD600 = 0,5-0,7). Клетки инфицировали фагом-помощником M1 и культивировали в среде, содержащей 2×YTCMK, 5 мМ MgCl2 и 1 мМ IPTG при 30°C в течение 16 ч. Культуры клеток центрифугировали (4500 об/мин, 15 мин, 4°С), а супернатанты переносили в новые пробирки (поли-scFv-фаги от каждого из трех пэннингов). На 96-луночных иммунопланшетах (NUNC 439454) фиксировали по два вида антигенов при плотности 100 нг на лунку в буфере для покрытия при 4°C в течение 16 часов, а затем каждую лунку блокировали раствором PBS с 4% обезжиренного молока.
Лунки промывали 0,2 мл PBS/T, в каждую лунку добавляли по 100 мкл поли-scFv-фагов от каждого из трех пэннингов и инкубировали при комнатной температуре в течение 2 ч. Каждую лунку снова промывали 4 раза по 0,2 мл PBS/T, добавляли вторичное антитело против M13-HRP (Amersham, 27-9421-01) в разведении 1:2000 и инкубировали при комнатной температуре в течение 1 часа. После промывки PBS/T растворяли таблетки OPD (Sigma, 8787-TAB) в буфере PC и полученный раствор добавляли в лунки по 100 мкл на лунку, индуцируя развитие окраски в течение 10 мин. Затем измеряли поглощение при 490 нм на спектрофотометре (Molecular Devices).
Результаты представлены на фиг. 3. Как видно из фиг. 3, анализ по ELISA показал, что у третьих поли-scFv-фагов повышается способность к связыванию с двумя антигенами PD-L1.
4. Скрининг положительных фагов
Колонии, полученные из группы поликлональных фаговых антител (из третьего пэннинга) с высокой способностью к связыванию, культивировали в 96-луночном планшете с глубокими лунками на 1 мл (Bioneer, 90030) при 37°C в течение 16 часов. По 100-200 мкл выращенных при этом клеток вносили в среду, содержащую 1 мл 2×YTCM, 2% глюкозы и 5 мМ MgCl2, до значения OD600 = 0,1, а затем культивировали в глубоком 96-луночном планшете при 37°C в течение 2-3 часов до OD600 = 0,5-0,7. Клетки инфицировали фагом-помощником M1 при MOI 1:20 и культивировали в среде, содержащей 2×YTCMK, 5 мМ MgCl2, 1 мМ IPTG при 30°C в течение 16 часов.
На 96-луночных иммунопланшетах фиксировали антиген PD-L1 при плотности 100 нг на лунку при 4°C в течение 16 ч, а затем каждую лунку блокировали раствором PBS с 4% обезжиренного молока. В каждую лунку вносили по 100 мкл моноклонального scFv-фага (100 scFv-фагов) после промывки 0,2 мл PBS/T и культивирования в течение 16 часов и инкубировали при комнатной температуре в течение 2 ч. Лунки опять промывали 4 раза по 0,2 мл PBS/T, добавляли вторичное антитело против M13-HRP в разведении 1:2000 и инкубировали при комнатной температуре в течение 1 часа. После промывки 0,2 мл PBS/T проводили развитие окраски и измеряли поглощение при 490 нм.
В результате, как видно из фиг. 4, было получено в общей сложности несколько десятков одиночных фаговых клонов для PD-L1 в виде моноклонов с высокой способностью к связыванию с каждым антигеном.
5. Анализ нуклеотидных последовательностей положительных фаговых антител
Из отобранных одиночных клонов выделяли ДНК с помощью набора для очистки ДНК (Qiagen, Germany) и заказывали анализ последовательностей этих ДНК (Solgent). Исходя из результатов анализа последовательностей, идентифицировали участки CDR из VH и VL у выбранных антител и исследовали сходство (идентичность) между этими антителами и группами гаметных антител с помощью программы Ig BLAST на веб-сайте NCBI по адресу http://www.ncbi.nlm.nih.gov/igblast/. В результате было получено 10 видов фаговых антител, специфичных к PD-L1, которые приведены ниже в таблице 3.
Таблица 3. Характеристики моноклонов PD-L1
клона
Антитела, включая последовательности участков CDR и FR тяжелой и легкой цепи выбранных антител, а также содержащие их вариабельные области тяжелой цепи и вариабельные области легкой цепи, представлены ниже в таблицах 4 и 5.
Таблица 4. Вариабельные области тяжелой цепи у клонов PD-L1
Таблица 5. Вариабельные области легкой цепи у клонов PD-L1
Пример 3. Получение человеческих антител к PD-L1
1. Преобразование из формата scFv в формат IgG
Для перевода выбранных 10 видов моноклональных фаговых антител к PD-L1 из фагов в целый IgG-вектор проводили ПЦР (iCycler iQ, BioRad) на тяжелых и легких цепях. В результате получали тяжелые и легкие цепи, а векторы и тяжелые и легкие цепи каждого из клонов разрезали (расщепляли) рестрикционными ферментами. Из каждого вектора и тяжелой цепи выделяли ДНК с помощью набора для экстракции ДНК из геля (Qiagen). Проводили лигирование путем смешивания 1 мкл (10 нг) вектора, 15 мкл (100-200 нг) тяжелой цепи, 2 мкл 10-кратного буфера, 1 мкл лигазы (1 ед./мкл) и дистиллированной воды, а затем инкубации смеси при комнатной температуре в течение 1-2 часов. Полученную смесь вводили в трансформируемые клетки (компетентные клетки, XL1-blue), помещали клетки на лед на 5 мин и подвергали их тепловому шоку при 42°C в течение 90 сек.
После теплового шока к клеткам добавляли 1 мл среды, а затем культивировали клетки при 37°C в течение 1 часа, рассеивали шпателем по чашке с LB Amp и инкубировали при 37°C в течение 16 часов. Полученные при этом колонии инокулировали в 5 мл среды LB Amp, культивировали при 37°C в течение 16 часов и проводили выделение ДНК с помощью набора DNA-prep (Nuclogen). Заказывали анализ последовательностей полученных ДНК (Solgent).
В результате было подтверждено, что последовательности тяжелых цепей и легких цепей у 11 клонов для PD-L1, переведенных в целые IgG, соответствуют последовательностям фаговых антител. Для трансфекции в клетки HEK293F тяжелые и легкие цепи соответствующих клонов, переведенных в целые IgG, культивировали в 100 мл среды LB Amp и выделяли ДНК с помощью набора Midi-prep (Qiagen).
2. Получение человеческих антител
Клонированные векторы pNATVH и pNATVL в соотношении 6:4 совместно трансфецировали в клетки HEK293F, на 7-й день собирали супернатанты, удаляли клетки и обломки клеток центрифугированием и через фильтры на 0,22 мкм, а супернатанты собирали и подвергали аффинной хроматографии на белке A для очистки IgG-антител. Антитела после очистки проводили через глициновый буфер и заменяли буфер таким образом, чтобы конечным буфером при ресуспендировании был PBS. Проводили количественное определение очищенных антител методом BCA и Nanodrop, а затем каждое из 10 видов антител наносили в дозе 5 мкг в восстановительных и невосстановительных условиях и анализировали методом SDS-PAGE для определения чистоты и подвижности очищенного белка (фиг. 5).
В результате, как видно из фиг. 5, в невосстановительных условиях все 10 антител детектировались при молекулярной массе 150 кДа и больше.
Пример 4. Характеристики моноклональных антител к PD-L1
1. Оценка активности антител
Проводили тестирование выбранных антител на активность с помощью набора для биоанализа блокировки PD1/PD-L1 (Promega, J1250). В 96-луночный планшет высеивали клетки линии CHO с высокой экспрессией PD-L1, культивировали в течение 16 часов или дольше, обрабатывали каждым антителом в серийном разведении при постоянной концентрации, а затем культивировали вместе с клетками линии Jurkat с высокой экспрессией PD-1 человека в течение 6 часов. Определяли степень восстановления ингибирования у антител на спектрофотометре (SpectraMax M5, Molecular Devices, USA) по интенсивности люминесценции, возникающей при расщеплении субстрата люциферазой. Активность 10 видов антител к PD-L1 определяли по значениям для восстановления пониженного сигнала при образовании комплекса PD-1/PD-L1, причем 16E12 проявляло активность, близкую контрольному антителу (фиг. 6).
Для измерения активности антитела 16E12 к PD-L1 в зависимости от концентрации делали серийные разведения и опять проводили биоанализ блокирования PD1/PD-L1 по восстановлению пониженного сигнала зависимым от концентрации образом. Степень восстановления выражали в виде EC50 (эффективная концентрация mAb при 50%-м уровне восстановления сигнала) при анализе с помощью Graphpad Prism6, а значения EC50 по способности к восстановлению ингибирования in vitro представлены на фиг. 7.
2. Сродство антитела к PD-L1 к гиперэкспрессированным клеткам
Обращаясь к пулам трансформированных клеток с высокой экспрессией PD-L1, клетки HEK293E трансформировали плазмидой pcDNA3.1, содержащей PD-L1 человека, и проводили скрининг в селективной среде, содержащей 150 мкг/мл зеоцина (#R25001, Thermo Fisher). Каждый пул клеток идентифицировали и отбирали методом активируемой флуоресценцией сортировки клеток (FACS) с помощью антитела против PD-L1 и использовали для таких функциональных анализов, как анализ связывания по FACS или анализ конкуренции по FACS.
Из пулов трансформированных клеток с высокой экспрессией PD-L1 человека отбирали образцы по 0,5-1×106 клеток, готовили серийные разведения антител при постоянной степени разведения и проводили реакцию с полученными клетками при 4°C в течение 20 мин. Затем клетки трижды промывали PBS (#LB001-02, Welgene), содержащим 2% фетальной телячьей сыворотки, и проводили реакцию при 4°C в течение 20 мин с антителом против IgG человека (#FI-3000, Vectorlabs), конъюгированным с флуоресцентным веществом FITC (флуоресцеин-изотиоцианат). Клетки опять промывали так же, как описано выше, а затем суспендировали в 0,5 мл PBS, содержащем 2% FBS (#26140-079, Thermo Fisher), и проводили проточную цитометрию на проточном цитометре FACSCanto II (BD Biosciences, USA). При этом антитело 16E12 к PD-L1 специфически связывалось, а его способность к связыванию определяли по константе равновесной диссоциации (Kd), полученной через функцию анализа Graphpad Prism6.
В результате, как видно из фиг. 8, способность к связыванию у антитела при зависимом от концентрации связывании с PD-L1 человека, гиперэкспрессированным на поверхности клеток, можно определять по MFI (средней интенсивности флуоресценции).
3. Сродство антитела к PD-L1 на приборе ProteOn XPR36
Использовали прибор ProteOn XPR36 (BioRad). На приборе устанавливали сенсорный чип GLC (BioRad) и промывали его буфером PBST, а поверхность карбоксиметилдекстрана активировали смесью растворов EDC и сульфо-NHS. На сенсорный чип GLC наносили и иммобилизировали PD-L1-hFc, растворенный при концентрации 5 мкг/мл в буферном растворе 10 мМ ацетата натрия (pH 5,0).
Для того чтобы дезактивировать активированные карбоксильные группы, не прореагировавшие с белком PD-L1, пропускали 1М этаноламин и вводили 10 мМ глицин (рН 2,0) с тем, чтобы смыть белки, не связавшиеся с сенсорным чипом. Затем регистрировали данные для сенсограммы при связывании и диссоциации по времени, пропуская антитела со скоростью подачи 30 мкл/мин (от 30 нМ до 0,123 нМ) в течение 10 мин с помощью буфера PBST.
Рассчитывали равновесную константу диссоциации (KD) путем нанесения на график и аппроксимации данных по сенсограмме в состоянии равновесия в зависимости от концентрации. При этом 16E12 проявляло значение KD 0,045 нМ, что указывает на высокое сродство к антигену PD-L1 (фиг. 9).
Пример 5. Оптимизация антитела 16E12 к PD-L1
1. Получение библиотек для оптимизации антитела 16E12 к PD-L1
Для оптимизации антитела получали новые библиотеки с перетасовкой LC путем иммобилизации тяжелой цепи и введения пула в 105-106 легких цепей (LC) от фирмы Ybiologics, Inc. Также проводили оптимизацию антитела следующими тремя способами: перетасовки LC; базовой упаковки + перетасовки LC, включая сравнительный анализ остатков в структурно важных сайтах типа гидрофобной сердцевины тяжелых цепей, открытых остатков, кластеров зарядов, солевых мостиков, мутационное превращение их в консервативные остатки, а затем проведение перетасовки LC; и горячих точек в CDR + перетасовки LC в случае ДНК вариабельных областей антител, включая случайный мутагенез мутационных горячих точек, которые могут часто мутировать в процессе созревания сродства in vivo, а затем проведение перетасовки LC.
Для получения библиотек с перетасовкой LC расщепляли гены LC антитела 16E12 с помощью BstXI и затем использовали их в качестве векторов, а библиотечные пулы фирмы Ybiologics, Inc. расщепляли с помощью BstXI и использовали в качестве вставок. После лигирования с помощью лигазы проводили трансформацию клеток методом электропорации. Библиотеки антител получали, собирая трансформированные клетки на квадратной пластине. При этом получали библиотеки с разнообразием в 1,5×107. Результаты анализа последовательностей показали, что все последовательности HC были идентичными, а последовательности LC отличались друг от друга.
Для получения библиотек типа базовой упаковки + перетасовки LC заменяли каркасные участки (FR) антитела 16E12 на последовательности из консервативных аминокислот, расщепляли гены LC с помощью BstXI и затем использовали их в качестве векторов, а библиотечные пулы фирмы Ybiologics, Inc. расщепляли с помощью BstXI и использовали в качестве вставок. После лигирования с помощью лигазы проводили трансформацию клеток методом электропорации. Библиотеки антител получали, собирая трансформированные клетки на квадратной пластине. При этом получали библиотеки с разнообразием в 8,4×106. Результаты анализа последовательностей показали, что участки FR в HC были заменены консервативными последовательностями аминокислот, а последовательности LC отличались друг от друга.
Для получения библиотек типа горячих точек CDR + перетасовки LC заменяли каркасные участки (FR) антитела 16E12 на последовательности из консервативных аминокислот, расщепляли библиотеки горячих точек CDR1 с помощью SfiI и использовали в качестве вставок, а библиотечные пулы фирмы Ybiologics, Inc. расщепляли с помощью SfiI и использовали в качестве векторов. После лигирования с помощью лигазы проводили трансформацию клеток методом электропорации. Библиотеки антител получали, собирая трансформированные клетки на квадратной пластине. При этом получали библиотеки с разнообразием в 5,6×106. Результаты анализа последовательностей показали, что участки FR в HC были заменены консервативными последовательностями аминокислот, горячие точки в CDR1 подверглись случайному мутагенезу, а последовательности LC отличались друг от друга.
Пример 6. Скрининг человеческих антител к PD-L1
1. Подготовка антигенов
В качестве белковых антигенов PD-L1-hFc и PD-L1-mFc, полученные на фирме Ybiologics, Inc, и PD-L1-his (кат. № 10377-H08H), приобретенный у Sino Biological Inc., фиксировали в дозе 50 мкг на иммуносорбционных пробирках, а затем блокировали.
2. Биопэннинг (просеивание)
Получали фаговую библиотеку человеческих антител путем инфицирования бактерий библиотекой scFv человека с разнообразием в 2,7×1010, а затем культивирования при 30°C в течение 16 ч. Культуральную среду после культивирования центрифугировали, а супернатант концентрировали с помощью PEG и затем растворяли в буфере PBS для получения библиотеки человеческих антител. Фаговую библиотеку человеческих антител вносили в иммунологическую пробирку и инкубировали при комнатной температуре в течение 2 часов. После отмывки 1 раз PBS/T и 1 раз PBS элюировались только scFv-фаги, специфически связавшиеся с антигеном.
Элюированными фагами опять инфицировали E. coli и проводили амплификацию (процесс пэннинга), получая пул положительных фагов. Для оптимизации антител проводили только первый раунд пэннинга. В результате, как видно из таблицы 7, оказалось, что количество фагов, связавшихся с антигеном (на выходе) при первом раунде пэннинга, немного повысилось по сравнению с количеством фагов на входе.
Таблица 6. Сравнение титра антител при оптимизационном пэннинге
3. Скрининг положительных фагов
Колонии, полученные при пэннинге, культивировали в 96-луночном планшете с глубокими лунками на 1 мл (Bioneer, 90030) при 37°C в течение 16 часов. По 100-200 мкл выращенных при этом клеток вносили в среду, содержащую 1 мл 2×YTCM, 2% глюкозы и 5 мМ MgCl2, до значения OD600 = 0,1, а затем культивировали в глубоком 96-луночном планшете при 37°C в течение 2-3 часов до OD600 = 0,5-0,7. Клетки инфицировали фагом-помощником M1 при MOI 1:20 и культивировали в среде, содержащей 2×YTCMK, 5 мМ MgCl2, 1 мМ IPTG при 30°C в течение 16 часов.
На 96-луночных иммунопланшетах фиксировали антиген PD-L1 при плотности 100 нг на лунку при 4°C в течение 16 ч, а затем каждую лунку блокировали раствором PBS с 4% обезжиренного молока. В каждую лунку вносили по 1 мкл моноклонального scFv-фага (100 scFv-фагов) после промывки 0,2 мл PBS/T и культивирования в течение 16 часов и инкубировали при комнатной температуре в течение 2 ч. Лунки опять промывали 4 раза по 0,2 мл PBS/T, добавляли вторичное антитело против M13-HRP в разведении 1:2000 и инкубировали при комнатной температуре в течение 1 часа. После промывки 0,2 мл PBS/T проводили развитие окраски и измеряли поглощение при 490 нм.
В результате были получены одиночные фаговые клоны с более высокой способностью к связыванию с каждым антигеном, чем у исходного антитела (16E12, отмечено красным цветом, 6D), а результаты представлены на фиг. 10.
4. Анализ нуклеотидных последовательностей положительных фаговых антител
Из отобранных одиночных клонов выделяли ДНК с помощью набора для очистки ДНК (Qiagen, Germany) и заказывали анализ последовательностей этих ДНК (Solgent). Исходя из результатов анализа последовательностей, идентифицировали участки CDR из VH и VL у выбранных антител и исследовали сходство (идентичность) между этими антителами и группами гаметных антител с помощью программы Ig BLAST на веб-сайте NCBI по адресу http://www.ncbi.nlm.nih.gov/igblast/. В результате был получен 21 вид фаговых антител с более высокой способностью к связыванию, чем у исходного антитела, а результаты приведены ниже в таблице 7.
Таблица 7. Характеристики оптимизированных моноклонов PD-L1
Антитела, включая последовательности участков CDR и FR тяжелой и легкой цепи выбранных антител, а также содержащие их вариабельные области тяжелой цепи и вариабельные области легкой цепи, представлены ниже в таблицах 8 и 9.
Таблица 8. Вариабельные области тяжелой цепи у человеческих антител к PD-L1
Таблица 9. Вариабельные области легкой цепи у человеческих антител к PD-L1
Пример 7. Получение человеческих антител к PD-L1
1. Преобразование из формата scFv в формат IgG
Для перевода выбранного 21 вида моноклональных фаговых антител к PD-L1 из фагов в целый IgG-вектор проводили ПЦР (iCycler iQ, BioRad) на тяжелых и легких цепях. В результате получали тяжелые и легкие цепи, а векторы и тяжелые и легкие цепи каждого из клонов разрезали (расщепляли) рестрикционными ферментами. Из каждого вектора и тяжелой цепи выделяли ДНК с помощью набора для экстракции ДНК из геля (Qiagen). Проводили лигирование путем смешивания 1 мкл (10 нг) вектора, 15 мкл (100-200 нг) тяжелой цепи, 2 мкл 10-кратного буфера, 1 мкл лигазы (1 ед./мкл) и дистиллированной воды, а затем инкубации смеси при комнатной температуре в течение 1-2 часов. Полученную смесь вводили в трансформируемые клетки (компетентные клетки, XL1-blue), помещали клетки на лед на 5 мин и подвергали их тепловому шоку при 42°C в течение 90 сек.
После теплового шока к клеткам добавляли 1 мл среды, а затем культивировали клетки при 37°C в течение 1 часа, рассеивали шпателем по чашке с LB Amp и инкубировали при 37°C в течение 16 часов. Полученные при этом колонии инокулировали в 5 мл среды LB Amp, культивировали при 37°C в течение 16 часов и проводили выделение ДНК с помощью набора DNA-prep (Nuclogen). Заказывали анализ последовательностей полученных ДНК (Solgent).
В результате было подтверждено, что последовательности тяжелых цепей и легких цепей у 21 клона для PD-L1, переведенных в целые IgG, соответствуют последовательностям фаговых антител. Для трансфекции в клетки HEK293F тяжелые и легкие цепи соответствующих клонов, переведенных в целые IgG, культивировали в 100 мл среды LB Amp и выделяли ДНК с помощью набора Midi-prep (Qiagen).
2. Получение человеческих антител
Клонированные векторы pNATVH и pNATVL в соотношении 6:4 совместно трансфецировали в клетки HEK293F, на 7-й день собирали супернатанты, удаляли клетки и обломки клеток центрифугированием и через фильтры на 0,22 мкм, а супернатанты собирали и подвергали аффинной хроматографии на белке A для очистки IgG-антител. Антитела после очистки проводили через глициновый буфер и заменяли буфер таким образом, чтобы конечным буфером при ресуспендировании был PBS. Проводили количественное определение очищенных антител методом BCA и Nanodrop, а затем каждое из 21 вида антител наносили в дозе 5 мкг в восстановительных и невосстановительных условиях и анализировали методом SDS-PAGE для определения чистоты и подвижности очищенного белка. Кроме того, некоторые супернатанты наносили на SDS-PAGE для сравнения скорости экспрессии с исходным антителом, причем большинство антител экспрессировались сильнее, чем исходное антитело.
Пример 8. Характеристики моноклональных антител к PD-L1
1. Оценка активности антител
Проводили тестирование выбранных антител на активность с помощью набора для биоанализа блокировки PD1/PD-L1 (Promega, J1250). В 96-луночный планшет высеивали клетки линии CHO с высокой экспрессией PD-L1, культивировали в течение 16 часов или дольше, обрабатывали каждым антителом в серийном разведении при постоянной концентрации, а затем культивировали вместе с клетками линии Jurkat с высокой экспрессией PD-1 человека в течение 6 часов. Определяли степень восстановления ингибирования у антител на спектрофотометре (SpectraMax M5, Molecular Devices, USA) по интенсивности люминесценции, возникающей при расщеплении субстрата люциферазой. Активность 21 вида антител к PD-L1 определяли по значениям для восстановления пониженного сигнала при образовании комплекса PD-1/PD-L1, причем антитела 4A7, 4A11, 4C9, 4F5, 4H5 и 4H8 проявляли большую активность, чем исходное антитело, близкую к активности контрольного антитела (фиг. 11 и таблица 10).
Таблица 10. Активность моноклонов от выбранных вариантов антител к PD-L1
Для измерения активности 6 видов антител к PD-L1 (4A7, 4A11, 4C9, 4F5, 4H5, 4H8) в зависимости от концентрации делали серийные разведения и опять проводили биоанализ блокирования PD1/PD-L1 по восстановлению пониженного сигнала зависимым от концентрации образом. Степень восстановления выражали в виде EC50 (эффективная концентрация mAb при 50%-м уровне восстановления сигнала) при анализе с помощью Graphpad Prism6, причем 4F5 проявляло самую высокую эффективность восстановления ингибирования in vitro по значению EC50 (фиг. 12).
2. Сродство антител к PD-L1 к гиперэкспрессированным клеткам
Что касается пулов трансформированных клеток с высокой экспрессией PD-L1, клетки HEK293E трансформировали плазмидой pcDNA3.1, содержащей PD-1 человека (NM_005018.2) или PD-L1 человека (NM_014143.2), и проводили скрининг в селективной среде, содержащей 400 мкг/мл зеоцина (#R25001, Thermo Fisher). Каждый пул клеток идентифицировали и отбирали методом активируемой флуоресценцией сортировки клеток (FACS) с помощью антитела против PD-1 (#557860, BD) и использовали для таких функциональных анализов, как анализ связывания по FACS или анализ конкуренции по FACS. Из пулов трансформированных клеток с высокой экспрессией PD-L1 человека отбирали образцы по 0,5-1×106 клеток, готовили серийные разведения антител при постоянной степени разведения и проводили реакцию с полученными клетками при 4°C в течение 20 мин. Затем клетки трижды промывали PBS (#LB001-02, Welgene), содержащим 2% фетальной телячьей сыворотки, и проводили реакцию при 4°C в течение 20 мин с антителом против IgG человека (#FI-3000, Vectorlabs), конъюгированным с флуоресцентным веществом FITC (флуоресцеин-изотиоцианат). Клетки опять промывали так же, как описано выше, а затем суспендировали в 0,5 мл PBS, содержащем 2% FBS (#26140-079, Thermo Fisher), и проводили проточную цитометрию на проточном цитометре FACSCanto II (BD Biosciences, USA) (таблица 11).
Таблица 11. Связывание отобранных вариантов антител к PD-L1 с PD-L1, экспрессированным на поверхности клеток
Из пулов трансформированных клеток с высокой экспрессией PD-L1 человека отбирали образцы по 0,5-1×106 клеток, готовили серийные разведения антител при постоянной степени разведения и проводили реакцию с полученными клетками при 4°C в течение 20 мин. Затем клетки трижды промывали PBS (#LB001-02, Welgene), содержащим 2% фетальной телячьей сыворотки, и проводили реакцию при 4°C в течение 20 мин с антителом против IgG человека (#FI-3000, Vectorlabs), конъюгированным с флуоресцентным веществом FITC (флуоресцеин-изотиоцианат). Клетки опять промывали так же, как описано выше, а затем суспендировали в 0,5 мл PBS, содержащем 2% FBS (#26140-079, Thermo Fisher), и проводили проточную цитометрию на проточном цитометре FACSCanto II (BD Biosciences, USA) (фиг. 13).
3. Способность антител ингибировать образование комплекса PD-1/PD-L1 при иммуноферментном анализе
В лунки 96-луночного иммунопланшета (#439454, Thermo) вносили PD-1-Fc человека (S1420, Y-Biologics), затем трижды промывали PBS, содержащим 0,05% Tween-20 (#P9416, Sigma-Aldrich), а затем еще с 4% обезжиренного молока (#232120, Becton, Dickinson and Company) и оставляли на 1 час при комнатной температуре для блокирования неспецифического связывания. В то же время проводили реакции PD-L1-His человека (S1479, Y-Biologics) с серийными разведениями антител при постоянной степени разведения при комнатной температуре в течение 1 часа, после чего оставляли в том же микропланшете при комнатной температуре на 1 час. Полученные продукты промывали так же, как и выше, в лунки микропланшета добавляли антитело против биотина-His (#MA1-21315-BTIN, Thermo) в разведении 1:2000 и инкубировали при комнатной температуре в течение 1 часа, затем добавляли антитело против стрептавидина с поли-HRP (#21140, Pierce) в разведении 1:5000 и инкубировали при комнатной температуре в течение 1 часа, а затем промывали таким же образом. К продуктам реакции добавляли 100 мкл раствора субстрата TMB (#T0440, Sigma-Aldrich), защищали от света и инкубировали при комнатной температуре в течение 3 мин. Добавляли 50 мкл 2,5 М серной кислоты (#S1478, Samchun) для остановки реакции и измеряли поглощение при 450 нм на спектрофотометре GM3000 (Glomax® Discover System, Promega). Результаты представлены на фиг. 14.
4. Сродство антител к PD-L1 на приборе ProteOn XPR36
Использовали прибор ProteOn XPR36 (BioRad). На приборе устанавливали сенсорный чип GLC (BioRad) и промывали буфером PBST, а поверхность карбоксиметилдекстрана активировали смесью растворов EDC и сульфо-NHS. На сенсорный чип GLC наносили и иммобилизировали PD-L1-hFc, растворенный при концентрации 5 мкг/мл в буферном растворе 10 мМ ацетата натрия (pH 5,0).
Для того, чтобы дезактивировать активированные карбоксильные группы, не прореагировавшие с белком PD-L1, пропускали 1М этаноламин и вводили 10 мМ глицин (рН 2,0) с тем, чтобы смыть белки, не связавшиеся с сенсорным чипом. Затем регистрировали данные для сенсограммы при связывании и диссоциации по времени, пропуская антитела со скоростью подачи 30 мкл/мин (от 30 нМ до 0,123 нМ) в течение 10 мин с помощью буфера PBST.
Рассчитывали равновесные константы диссоциации (KD) путем нанесения на график и аппроксимации данных по сенсограмме в состоянии равновесия в зависимости от концентрации. При этом 16E12(4F5) проявляло значение KD 0,001 нМ, что указывает на высокое сродство к антигену PD-L1 (фиг. 15).
Сравнение сродства PDL1-16E12, LS и 4F5 к белкам PD-L1 человека, обезьяны и мыши представлено в таблице 12.
Таблица 12. Способность выбранного моноклонального антитела к PD-L1 (16E12-4F5) к связыванию с PD-L1 человека, обезьяны и мыши
Пример 9. Определение эпитопов у моноклональных антител к PD-L1
На 96-луночных иммунопланшетах фиксировали антиген PD-L1 дикого типа (WT) или некоторые варианты при плотности 100 нг на лунку при 4°C в течение 16 ч, а затем блокировали лунки раствором PBS с 4% обезжиренного молока. Каждую лунку промывали 0,2 мл PBS/T, а затем вносили одиночный клон scFv-фага (по 100 scFv-фагов) в дозе 100 мкл после культивирования в течение 16 часов и инкубировали при комнатной температуре в течение 2 часов. Лунки опять промывали 4 раза по 0,2 мл PBS/T, добавляли второе антитело против Fab в разведении 1:2000 и инкубировали при комнатной температуре в течение 1 часа. После промывки 0,2 мл PBS/T проводили развитие окраски и измеряли поглощение при 490 нм.
В результате оказалось, что контрольное антитело к PD-L1 и мутанты проявляют различные характеристики связывания, следовательно, они содержат разные эпитопы (фиг. 16).
Пример 10. Повышение активности под действием моноклонального антитела к PD-L1 при аллогенной реакции MLR
Смешивали Т-клетки с происходящими из моноцитов дендритными клетками, выделенными от разных людей, в соотношении 1:10 и культивировали в течение 5 дней, а затем измеряли количество γ-интерферона в культуральной среде. При этом культуральные среды, содержащие исходное антитело 16E12, проявляли зависимое от концентрации повышение количества γ-интерферона (фиг. 17).
Пример 11. Оценка эффективности моноклонального антитела к PD-L1 на модели рака у сингенных животных
Для установления эффективности моноклонального антитела 16E12-2B9 к PD-L1 in vivo в бока мышей BALb/C вводили подкожно 8×106 клеток CT-26 в качестве клеток рака толстой кишки и наблюдали рост опухолей при введении моноклонального антитела в дозе 5 мг/кг два раза в неделю на протяжении 3 недель с того времени, когда опухоли по размеру были как просяное зерно. При этом в группе, получавшей моноклональное антитело к PD-L1, наблюдалось значительное уменьшение размера опухолей (фиг. 18).
Пример 12. Испытание моноклональных антител к PD-L1 на термостабильность
Белок антител разводили в DPBS до 3 мкМ, отбирали 45 мкл и смешивали с 5 мкл 200-кратного красителя Sypro Orange (#S6650, Thermo), а затем по 50 мкл вносили в пробирки для qPCR (#B77009, B57651, Bioplastics). Проводили количественный ПЦР (qPCR) в реальном времени на установке BioRad CFX96. Условия qPCR задавали следующим образом: реакция при 25°C в течение 30 сек, повышение температуры на 1°C вплоть до 99°C и в то же время реакция при каждой температуре в течение 1 мин, а затем заключительная реакция при 25°C в течение 10 сек. В качестве константы, характеризующей разупорядочение структуры антител, использовали Tm (температура плавления). Результаты представлены ниже в таблице 13.
Таблица 13. Термостабильность антител
Пример 13. Определение связывания с PD-L2
Для изучения связывания антител против PD-L1 с PD-L2 в лунках 96-луночного иммунопланшета (#439454, Thermo) иммобилизировали PD-L2-Fc человека (#10292-H02H, Sino), а затем трижды промывали PBS, содержащим 0,05% Tween-20 (#P9416, Sigma-Aldrich), после чего оставляли на 1 час при комнатной температуре в чистящем растворе, содержащем 4% обезжиренного молока (#232120, Becton, Dickinson and Company), для блокировки неспецифического связывания. В то же время проводили реакции серийных разведений антител при постоянной степени разведения или PD-1-His человека (S1352, Y-Biologics) в качестве положительного контроля при комнатной температуре в течение 1 часа, после чего оставляли в том же планшете при комнатной температуре на 1 час. Полученные продукты промывали так же, как и выше, в лунки планшета добавляли антитело против биотина-His (#MA1-21315-BTIN, Thermo) в разведении 1:2000 и инкубировали при комнатной температуре в течение 1 часа, затем добавляли антитело против стрептавидина с поли-HRP (#21140, Pierce) в разведении 1:5000 и инкубировали при комнатной температуре в течение 1 часа, а затем промывали таким же образом. К продуктам реакции добавляли 100 мкл раствора субстрата TMB (#T0440, Sigma-Aldrich), защищали от света и инкубировали при комнатной температуре в течение 3 мин. Добавляли 50 мкл 2,5 М серной кислоты (#S1478, Samchun) для остановки реакции и измеряли поглощение при 450 нм на спектрофотометре GM3000 (Glomax® Discover System, Promega). Результаты представлены на фиг. 19.
Промышленная применимость
Новые антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты по настоящему изобретению могут связываться с PD-L1 с высоким сродством и ингибировать образование комплекса PD-1/PD-L1, тем самым ингибируя истощение Т-клеток в обход опосредованной PD-1/PD-L1 активности Т-клеток. Соответственно, антитела, связывающиеся с PD-L1, либо их антигенсвязывающие фрагменты по настоящему изобретению применимы для профилактики или лечения целевых раковых либо инфекционных заболеваний.
Хотя конкретные конфигурации настоящего изобретения были описаны подробно, однако специалистам в данной области должно быть ясно, что данное описание приводится в качестве предпочтительных воплощений для иллюстративных целей и не должно рассматриваться как ограничивающее объем настоящего изобретения. Таким образом, существенный объем настоящего изобретения определяется прилагаемой формулой изобретения и ее эквивалентами.
--->
SEQUENCE LISTING
<110> Y-BIOLOGICS INC.
<120> Antibodies Against Programmed death-ligand 1 and Uses Thereof
<130> PF-B2150
<140> PCT/KR2017/008495
<141> 2017-08-07
<150> KR10-2016-0100211
<151> 2016-08-05
<160> 251
<170> PatentIn version 3.5
<210> 1
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 1
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 2
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 2
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 3
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 3
Gly Asp Thr Phe Ser Arg Tyr Ala
1 5
<210> 4
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 4
Gly Glu Asn Phe Arg Ser His Ala
1 5
<210> 5
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 5
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 6
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 6
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 7
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 7
Gly Phe Thr Phe Ser Ser Tyr Val
1 5
<210> 8
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 8
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 9
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 9
Ile Ile Pro Ile Leu Gly Ile Ala
1 5
<210> 10
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 10
Ile Ile Pro Val Leu Gly Ala Ala
1 5
<210> 11
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 11
Ile Ile Pro Ile Val Gly Leu Ala
1 5
<210> 12
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 12
Ile Ile Pro Met Phe Asp Met Thr
1 5
<210> 13
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 13
Ile Ser Tyr Asp Gly Ser Asn Glu
1 5
<210> 14
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 14
Ile Ser Ser Asn Gly Gly Ser Thr
1 5
<210> 15
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 15
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 16
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 16
Ala Arg Asp Pro Trp Gly Gly Tyr Tyr Gly Met Asp Val
1 5 10
<210> 17
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 17
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile
1 5 10
<210> 18
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 18
Ala Arg Asp Phe Val Leu Ser Gly Ser Ala Thr Val Phe Asp Pro
1 5 10 15
<210> 19
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 19
Ala Arg Ser Leu Ser Gly Tyr Ser Leu Gly Ala Phe Asp Val
1 5 10
<210> 20
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 20
Val Thr Gly Arg Pro Pro Trp His
1 5
<210> 21
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 21
Ala Arg Ser Asn Pro His Tyr Met Asp Val
1 5 10
<210> 22
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 22
Ala Arg Gly Asp Tyr Asn Gly Val Tyr Met Asp Val
1 5 10
<210> 23
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 23
Ala Arg Ala Ser Ser Gly Tyr Ser Val Gly Ala Phe Asp Ile
1 5 10
<210> 24
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 24
Ala Arg Glu His Phe Ser Thr Tyr Ser Tyr Gly Val Asp Val
1 5 10
<210> 25
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 25
Ala Arg Gly Gly Arg Ser Phe Gly Ala Phe Asp Ser
1 5 10
<210> 26
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 26
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser
20 25
<210> 27
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 27
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser
20 25
<210> 28
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 28
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Arg Ala Ser
20 25
<210> 29
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 29
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Val Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser
20 25
<210> 30
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 30
Gln Met Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser
20 25
<210> 31
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 31
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser
20 25
<210> 32
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 32
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser
20 25
<210> 33
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 33
Gln Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser
20 25
<210> 34
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 34
Gln Met Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ser Ala Ser
20 25
<210> 35
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 35
Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met Gly
1 5 10 15
Gly
<210> 36
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 36
Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly
1 5 10 15
Arg
<210> 37
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 37
Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Ala Pro Glu Trp Met Gly
1 5 10 15
Arg
<210> 38
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 38
Phe Ser Trp Val Arg Gln Ala Pro Gly His Gly Leu Glu Trp Met Gly
1 5 10 15
Arg
<210> 39
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 39
Met His Trp Val Arg Gln Gly Pro Gly Lys Gly Leu Glu Trp Val Ala
1 5 10 15
Val
<210> 40
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 40
Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Tyr Val Ser
1 5 10 15
Ala
<210> 41
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 41
Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met Gly
1 5 10 15
Gly
<210> 42
<211> 38
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 42
Ile Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Met Thr Glu Asp Thr
1 5 10 15
Ser Thr Asp Thr Ala Tyr Met Glu Leu Ser Ser Leu Arg Ser Glu Asp
20 25 30
Thr Ala Val Tyr Tyr Cys
35
<210> 43
<211> 38
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 43
Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Ile Thr Ala Asp Lys
1 5 10 15
Ser Thr Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Arg Ser Glu Asp
20 25 30
Thr Ala Val Tyr Tyr Cys
35
<210> 44
<211> 38
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 44
Asn Tyr Ala Arg Lys Phe Gln Asp Arg Val Thr Ile Thr Ala Asp Ile
1 5 10 15
Ser Thr Thr Thr Ala Phe Met Glu Leu Thr Ser Leu Arg Ser Glu Asp
20 25 30
Thr Ala Val Tyr Tyr Cys
35
<210> 45
<211> 38
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 45
Asp Tyr Pro Gln Lys Phe Gln Gly Arg Val Thr Ile Ser Ala Asp Glu
1 5 10 15
Ser Thr Gly Thr Ala Tyr Met Asp Leu Ser Ser Leu Arg Ser Glu Asp
20 25 30
Thr Ala Val Tyr Tyr Cys
35
<210> 46
<211> 38
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 46
Asp Tyr Ala Gln Arg Phe Gln Gly Arg Leu Thr Ile Ile Ala Asp Glu
1 5 10 15
Ser Ser Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Arg Ser Glu Asp
20 25 30
Thr Ala Val Tyr Tyr Cys
35
<210> 47
<211> 38
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 47
Phe Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
1 5 10 15
Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp
20 25 30
Thr Ala Val Tyr Tyr Cys
35
<210> 48
<211> 38
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 48
Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
1 5 10 15
Ser Lys Asn Thr Leu Tyr Leu Gln Met Ser Ser Leu Arg Ser Glu Asp
20 25 30
Thr Ala Val Tyr Tyr Cys
35
<210> 49
<211> 38
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 49
Asn Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Ile Thr Ala Asp Glu
1 5 10 15
Ser Thr Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Arg Ser Glu Asp
20 25 30
Thr Ala Val Tyr Tyr Cys
35
<210> 50
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 50
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
1 5 10
<210> 51
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 51
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
1 5 10
<210> 52
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 52
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 53
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 53
Trp Gly Pro Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 54
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 54
Trp Gly Gln Gly Ile Leu Ile Thr Val Ser Ser
1 5 10
<210> 55
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 55
Trp Gly Pro Gly Thr Thr Val Thr Val Ser Ser
1 5 10
<210> 56
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 56
Trp Gly Gln Gly Ala Leu Val Thr Val Ser Ser
1 5 10
<210> 57
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 57
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Pro Trp Gly Gly Tyr Tyr Gly Met Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 58
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 58
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 59
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 59
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Arg Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Phe Val Leu Ser Gly Ser Ala Thr Val Phe Asp Pro Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 60
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 60
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Val Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Asp Thr Phe Ser Arg Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Ala Pro Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Val Leu Gly Ala Ala Asn Tyr Ala Arg Lys Phe
50 55 60
Gln Asp Arg Val Thr Ile Thr Ala Asp Ile Ser Thr Thr Thr Ala Phe
65 70 75 80
Met Glu Leu Thr Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Leu Ser Gly Tyr Ser Leu Gly Ala Phe Asp Val Trp Gly
100 105 110
Pro Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 61
<211> 115
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 61
Gln Met Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Glu Asn Phe Arg Ser His
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Val Gly Leu Ala Asp Tyr Pro Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Ser Ala Asp Glu Ser Thr Gly Thr Ala Tyr
65 70 75 80
Met Asp Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Thr Gly Arg Pro Pro Trp His Trp Gly Gln Gly Ile Leu Ile Thr
100 105 110
Val Ser Ser
115
<210> 62
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 62
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Phe Ser Trp Val Arg Gln Ala Pro Gly His Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Met Phe Asp Met Thr Asp Tyr Ala Gln Arg Phe
50 55 60
Gln Gly Arg Leu Thr Ile Ile Ala Asp Glu Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Asn Pro His Tyr Met Asp Val Trp Gly Gln Gly Thr Thr
100 105 110
Val Thr Val Ser Ser
115
<210> 63
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 63
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Gly Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Glu Phe Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Asp Tyr Asn Gly Val Tyr Met Asp Val Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210> 64
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 64
Gln Val Gln Leu Val Glu Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Ser Ser Gly Tyr Ser Val Gly Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 65
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 65
Gln Met Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ser Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Tyr Val
35 40 45
Ser Ala Ile Ser Ser Asn Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu His Phe Ser Thr Tyr Ser Tyr Gly Val Asp Val Trp Gly
100 105 110
Pro Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 66
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 66
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Gly Arg Ser Phe Gly Ala Phe Asp Ser Trp Gly Gln Gly
100 105 110
Ala Leu Val Thr Val Ser Ser
115
<210> 67
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 67
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 68
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 68
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 69
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 69
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 70
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 70
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 71
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 71
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 72
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 72
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 73
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 73
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 74
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 74
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 75
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 75
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 76
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 76
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 77
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 77
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 78
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 78
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 79
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 79
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 80
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 80
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 81
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 81
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 82
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 82
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 83
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 83
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 84
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 84
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 85
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 85
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 86
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 86
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 87
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 87
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Arg Ile Ile Pro Ile Leu Gly Ile Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Pro Arg Asp Gly Tyr Asn Leu Val Ala Phe Asp Ile Trp Gly
100 105 110
Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 88
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 88
Gln Gly Ile Arg Thr Trp
1 5
<210> 89
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 89
Ser Ser Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 90
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 90
Ser Thr Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 91
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 91
Asn Ser Asn Ile Gly Thr Pro Tyr Asp
1 5
<210> 92
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 92
Asn Ser Asp Ile Gly Ala Gly His Asp
1 5
<210> 93
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 93
Gln Ser Leu Leu His Ser Asp Gly Lys Thr Tyr
1 5 10
<210> 94
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 94
Ser Ser Asn Ile Gly Asn Asn Tyr
1 5
<210> 95
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 95
Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr
1 5 10
<210> 96
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 96
Gln Ser Ile Ser Ser Tyr
1 5
<210> 97
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 97
Asn Ser Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 98
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 98
Asn Ser Asn Ile Gly Ala Gly His Asp
1 5
<210> 99
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 99
Ser Ser Asn Leu Gly Ala Pro Tyr Asp
1 5
<210> 100
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 100
Ser Ser Asn Ile Gly Ala Gly Ser Asp
1 5
<210> 101
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 101
Arg Ser Asn Phe Gly Ala Gly His Asp
1 5
<210> 102
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 102
Ser Ser Asn Val Gly Ala Gly Tyr Asp
1 5
<210> 103
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 103
Ala Ala Ser
1
<210> 104
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 104
Gly Asn Asn
1
<210> 105
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 105
Gly Asn Ser
1
<210> 106
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 106
Gly Ser Thr
1
<210> 107
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 107
Ser Asn Arg
1
<210> 108
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 108
Gly Asn Thr
1
<210> 109
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 109
Glu Val Ser
1
<210> 110
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 110
Asp Asn Asn
1
<210> 111
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 111
Met Ile Ser
1
<210> 112
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 112
Asp Ser Ser
1
<210> 113
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 113
Gly Asp Asn
1
<210> 114
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 114
Thr Asn His
1
<210> 115
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 115
Gly Asp Arg
1
<210> 116
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 116
Gly Asp Thr
1
<210> 117
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 117
Glu Asn Ser
1
<210> 118
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 118
Ala Asn Thr
1
<210> 119
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 119
Gly Asp Ile
1
<210> 120
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 120
Gln Gln Ala Asn Ser Phe Pro Leu Thr
1 5
<210> 121
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 121
Gln Ser Tyr Asp Asn Ser Leu Ser Ala His Ala Val
1 5 10
<210> 122
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 122
Gln Ser Tyr Asp Ser Ser Leu Ser Gly Tyr Val
1 5 10
<210> 123
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 123
Gln Ser Tyr Asp Arg Ser Leu Gly Val Ser Asp Val Val
1 5 10
<210> 124
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 124
Gln Ser Tyr Asp Ser Ser Leu Ser Gly Pro Asn Tyr Val
1 5 10
<210> 125
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 125
Met Gln Gly Leu His Leu Pro Tyr Thr
1 5
<210> 126
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 126
Gly Thr Trp Asp Ser Ser Leu Ser Ala Gly Val
1 5 10
<210> 127
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 127
Met Gln Arg Val Glu Phe Pro Tyr Thr
1 5
<210> 128
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 128
Gln Gln Arg Gly Gln Trp Pro Leu Thr
1 5
<210> 129
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 129
Gln Ser Phe Asp Arg Ser Arg Ser Gly Ser Asn Val
1 5 10
<210> 130
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 130
His Ser Tyr Asp Gly Ser Leu Ser Glu Gly Val
1 5 10
<210> 131
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 131
Gln Ser Phe Asp Ser Arg Leu Gly Val Val
1 5 10
<210> 132
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 132
Gln Ser Tyr Asp Asn Ser Leu Arg Gly Ser Val
1 5 10
<210> 133
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 133
Gln Ser Tyr Asp Asn Arg Leu Ser Gly Ser Leu
1 5 10
<210> 134
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 134
Gln Ser Tyr Asp Ser Arg Leu Ser Gly Pro Val
1 5 10
<210> 135
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 135
Gln Ser Tyr Asn Asn Ser Leu Arg Gly Ser Val
1 5 10
<210> 136
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 136
Gln Ser Tyr Asp Ser Ser Leu Gly Gly Ser Val
1 5 10
<210> 137
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 137
Gln Ser Tyr Asp Ser Ser Leu Ser Ser Ser Val
1 5 10
<210> 138
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 138
Gln Ser Tyr Asp Ser Arg Leu Gly Ala Val
1 5 10
<210> 139
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 139
Gln Ser Tyr Asp Ser Ser Leu Ser Gly Gly Val
1 5 10
<210> 140
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 140
Gln Ser Tyr Asp Ser Ser Leu Asn Gly Val Val
1 5 10
<210> 141
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 141
Gln Ser Tyr Asp Ser Ser Leu Ser Gly Ser Val
1 5 10
<210> 142
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 142
Gln Ser Tyr Asp Ser Ser Leu Arg Val Ser Ser Val
1 5 10
<210> 143
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 143
Gln Ser Tyr Asp Ser Ser Leu Ser Gly Ser Gly Val
1 5 10
<210> 144
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 144
Gln Ser Tyr Asp Ser Arg Leu Gly Val Val
1 5 10
<210> 145
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 145
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser
20 25
<210> 146
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 146
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Thr Val Thr Ile Ser Cys Thr Gly Ser
20 25
<210> 147
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 147
Gln Phe Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser
20 25
<210> 148
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 148
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Ser Val Ser Ile Ser Cys Ile Gly Ser
20 25
<210> 149
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 149
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser
20 25
<210> 150
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 150
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Ser Val Thr Pro Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser
20 25
<210> 151
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 151
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Ala Pro Pro Gly Gln
1 5 10 15
Lys Val Thr Ile Ser Cys Ser Gly Ser
20 25
<210> 152
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 152
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser
20 25
<210> 153
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 153
Asp Ile Gln Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Thr Ala Ser
20 25
<210> 154
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 154
Gln Leu Val Leu Thr Gln Pro Thr Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Ile Ile Ser Cys Thr Gly Ser
20 25
<210> 155
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 155
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Ile Ile Ser Cys Thr Gly Ser
20 25
<210> 156
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> ARTIFICIAL SEQUENCE
<400> 156
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Thr
20 25
<210> 157
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 157
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Thr
20 25
<210> 158
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 158
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Ser Val Thr Ile Ser Cys Thr Gly Ser
20 25
<210> 159
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 159
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Gly
20 25
<210> 160
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 160
Gln Leu Val Leu Thr Gln Pro Ser Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser
20 25
<210> 161
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 161
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Gly Val Thr Ile Ser Cys Thr Gly Asp
20 25
<210> 162
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 162
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Lys Val Thr Phe Ser Cys Thr Gly Ser
20 25
<210> 163
<211> 25
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 163
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Thr Val Thr Ile Ser Cys Thr Gly Ser
20 25
<210> 164
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 164
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
1 5 10 15
Tyr
<210> 165
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 165
Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile
1 5 10 15
Tyr
<210> 166
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 166
Val His Trp Tyr Gln Gln Leu Pro Gly Lys Ala Pro Lys Val Val Ile
1 5 10 15
Tyr
<210> 167
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 167
Val His Trp Tyr Gln Gln Ile Pro Gly Glu Ala Pro Lys Leu Leu Ile
1 5 10 15
Tyr
<210> 168
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 168
Val His Trp Tyr Gln Gln Ile Pro Gly Thr Ala Pro Lys Val Leu Ile
1 5 10 15
Tyr
<210> 169
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 169
Leu Tyr Trp Tyr Leu Gln Lys Pro Gly Gln Ser Pro Gln Leu Leu Ile
1 5 10 15
Tyr
<210> 170
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 170
Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile
1 5 10 15
Tyr
<210> 171
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 171
Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser Pro Gln Leu Leu Ile
1 5 10 15
Tyr
<210> 172
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 172
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
1 5 10 15
Tyr
<210> 173
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 173
Val His Trp Tyr Gln Ser Leu Pro Gly Thr Val Pro Lys Leu Leu Ile
1 5 10 15
Ser
<210> 174
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 174
Val His Trp Tyr Gln His Arg Pro Gly Lys Ala Pro Gln Leu Leu Ile
1 5 10 15
Tyr
<210> 175
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 175
Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile
1 5 10 15
His
<210> 176
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 176
Val His Trp Tyr Gln Gln Val Pro Gly Thr Ala Pro Lys Leu Leu Ile
1 5 10 15
Tyr
<210> 177
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 177
Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile
1 5 10 15
Val
<210> 178
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 178
Val His Trp Tyr Gln Gln Leu Pro Gly Ala Ala Pro Lys Leu Leu Ile
1 5 10 15
His
<210> 179
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 179
Val Gln Trp Tyr Gln Gln Ile Pro Gly Thr Ala Pro Lys Leu Leu Ile
1 5 10 15
Tyr
<210> 180
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 180
Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Ile Ile
1 5 10 15
Tyr
<210> 181
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 181
Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Leu
1 5 10 15
Phe
<210> 182
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 182
Val His Trp Tyr Gln Gln Phe Pro Gly Thr Ala Pro Lys Leu Leu Ile
1 5 10 15
Tyr
<210> 183
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 183
Val His Trp Tyr Gln His Leu Pro Gly Ala Ala Pro Arg Leu Leu Ile
1 5 10 15
Tyr
<210> 184
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 184
Val His Trp Tyr Gln Gln Leu Pro Gly Ala Ala Pro Lys Leu Leu Ile
1 5 10 15
Tyr
<210> 185
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 185
Asn Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly
1 5 10 15
Thr Asp Phe Thr Leu Thr Ile Ala Ser Leu Gln Pro Glu Asp Phe Ala
20 25 30
Thr Tyr Tyr Cys
35
<210> 186
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 186
Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Ala Leu Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Gly Ala
20 25 30
Asp Tyr His Cys
35
<210> 187
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 187
Asn Arg Pro Ser Gly Val Leu Asp Arg Phe Ser Ala Ser Lys Ser Ala
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 188
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 188
Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Arg Ser Gly
1 5 10 15
Ser Ser Ala Ser Leu Asp Ile Thr Gly Leu Gln Ala Asp Asp Glu Ala
20 25 30
Tyr Tyr Tyr Cys
35
<210> 189
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 189
Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 190
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 190
Asp Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 191
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 191
Lys Arg Phe Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
1 5 10 15
Thr Asp Phe Thr Leu Lys Ile Gly Arg Val Glu Ala Glu Asp Val Gly
20 25 30
Val Tyr Tyr Cys
35
<210> 192
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 192
Glu Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Ala Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Thr Leu Gly Ile Ser Gly Leu Gln Thr Gly Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 193
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 193
Phe Arg Ala Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly
1 5 10 15
Thr Asp Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Ser Gly
20 25 30
Ile Tyr Tyr Cys
35
<210> 194
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 194
Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly Ser Gly Ser Gly
1 5 10 15
Thr Ala Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu Asp Phe Ala
20 25 30
Val Tyr Phe Cys
35
<210> 195
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 195
Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ala Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 196
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 196
Asn Arg Pro Pro Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Ser Leu Gln Ser Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 197
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 197
Asn Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Pro Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 198
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 198
Ile Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Arg Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Asp Asp Glu Ala
20 25 30
Asn Tyr Phe Cys
35
<210> 199
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 199
His Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Glu Thr
20 25 30
Asp Tyr Tyr Cys
35
<210> 200
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 200
Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Asp
1 5 10 15
Thr Ser Ala Ser Leu Ala Leu Thr Gly Leu Gln Ala Glu Asp Glu Ala
20 25 30
Thr Tyr Tyr Cys
35
<210> 201
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 201
Asp Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Pro Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 202
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 202
Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Leu Ala Ser Leu Ala Ile Thr Gly Leu Gln Ser Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 203
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 203
Lys Arg Ala Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Gly Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 204
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 204
Asn Arg Pro Ser Gly Val Pro Gly Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Ser Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ser Asp Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 205
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 205
Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Val Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 206
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 206
Asn Arg Pro Leu Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 207
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 207
Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Ile Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 208
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 208
Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Thr Asp Asp Glu Ala
20 25 30
Asp Tyr Phe Cys
35
<210> 209
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 209
Asn Arg Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Ala Gly Leu Gln Ser Asp Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 210
<211> 36
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 210
Asn Arg Pro Ser Gly Val Pro Ala Arg Phe Ser Gly Ser Lys Ser Gly
1 5 10 15
Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu Gln Ala Glu Asp Glu Ala
20 25 30
Asp Tyr Tyr Cys
35
<210> 211
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 211
Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
1 5 10
<210> 212
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 212
Phe Gly Thr Gly Thr Lys Val Thr Val Leu
1 5 10
<210> 213
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 213
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
1 5 10
<210> 214
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 214
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
1 5 10
<210> 215
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 215
Phe Gly Gly Gly Thr Lys Val Asp Ile Lys
1 5 10
<210> 216
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 216
Ile Gly Thr Gly Thr Lys Val Thr Val Leu
1 5 10
<210> 217
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 217
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Val Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Gly Ile Arg Thr Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Asn Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ala Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ala Asn Ser Phe Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 218
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 218
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Thr Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Asn Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Ala Leu Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Gly Ala Asp Tyr His Cys Gln Ser Tyr Asp Asn Ser
85 90 95
Leu Ser Ala His Ala Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 219
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 219
Gln Phe Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Thr Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Lys Ala Pro Lys Val
35 40 45
Val Ile Tyr Gly Asn Ser Asn Arg Pro Ser Gly Val Leu Asp Arg Phe
50 55 60
Ser Ala Ser Lys Ser Ala Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Gly Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 220
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 220
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Ser Val Ser Ile Ser Cys Ile Gly Ser Asn Ser Asn Ile Gly Thr Pro
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Ile Pro Gly Glu Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Ser Thr Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Arg Ser Gly Ser Ser Ala Ser Leu Asp Ile Thr Gly Leu
65 70 75 80
Gln Ala Asp Asp Glu Ala Tyr Tyr Tyr Cys Gln Ser Tyr Asp Arg Ser
85 90 95
Leu Gly Val Ser Asp Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val
100 105 110
Leu
<210> 221
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 221
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Ser Asn Arg Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Gly Pro Asn Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val
100 105 110
Leu
<210> 222
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 222
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Asn Ser Asp Ile Gly Ala Gly
20 25 30
His Asp Val His Trp Tyr Gln Gln Ile Pro Gly Thr Ala Pro Lys Val
35 40 45
Leu Ile Tyr Gly Asn Thr Asp Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Gly Tyr Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 223
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 223
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Ser Val Thr Pro Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asp Gly Lys Thr Tyr Leu Tyr Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Glu Val Ser Lys Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Gly Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Gly
85 90 95
Leu His Leu Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 224
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 224
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Ala Pro Pro Gly Gln
1 5 10 15
Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Asp Asn Asn Glu Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Ala Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Ser Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
85 90 95
Ser Ala Gly Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 225
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 225
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Asp Ser
20 25 30
Asp Asp Gly Asn Thr Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln
35 40 45
Ser Pro Gln Leu Leu Ile Tyr Met Ile Ser Phe Arg Ala Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys
65 70 75 80
Ile Ser Arg Val Glu Ala Glu Asp Ser Gly Ile Tyr Tyr Cys Met Gln
85 90 95
Arg Val Glu Phe Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile
100 105 110
Lys
<210> 226
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 226
Asp Ile Gln Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Thr Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ser Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Ala Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Arg Gly Gln Trp Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Asp Ile Lys
100 105
<210> 227
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 227
Gln Leu Val Leu Thr Gln Pro Thr Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Ile Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Thr Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ala Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Phe Asp Arg Ser
85 90 95
Arg Ser Gly Ser Asn Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 228
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 228
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Ser Leu Pro Gly Thr Val Pro Lys Leu
35 40 45
Leu Ile Ser Gly Asn Ser Asn Arg Pro Pro Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Ser Leu
65 70 75 80
Gln Ser Glu Asp Glu Ala Asp Tyr Tyr Cys His Ser Tyr Asp Gly Ser
85 90 95
Leu Ser Glu Gly Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 229
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 229
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln His Arg Pro Gly Lys Ala Pro Gln Leu
35 40 45
Leu Ile Tyr Gly Asp Asn Asn Arg Pro Ser Gly Ile Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Phe Asp Ser Arg
85 90 95
Leu Gly Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 230
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 230
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Asn Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile His Gly Asn Asn Ile Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Arg Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Asp Asp Glu Ala Asn Tyr Phe Cys Gln Ser Tyr Asp Asn Ser
85 90 95
Leu Arg Gly Ser Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 231
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 231
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Ile Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Val Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Asn His Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Thr Asp Tyr Tyr Cys Gln Ser Tyr Asp Asn Arg
85 90 95
Leu Ser Gly Ser Leu Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 232
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 232
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Thr Asn Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Val Thr Asn His Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Asp Thr Ser Ala Ser Leu Ala Leu Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Thr Tyr Tyr Cys Gln Ser Tyr Asp Ser Arg
85 90 95
Leu Ser Gly Pro Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 233
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 233
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Ser Val Thr Ile Ser Cys Thr Gly Ser Asn Ser Asp Ile Gly Ala Gly
20 25 30
His Asp Val His Trp Tyr Gln Gln Leu Pro Gly Ala Ala Pro Lys Leu
35 40 45
Leu Ile His Gly Asp Arg Asp Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asn Asn Ser
85 90 95
Leu Arg Gly Ser Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 234
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 234
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Thr Asn Ser Asn Ile Gly Ala Gly
20 25 30
His Asp Val Gln Trp Tyr Gln Gln Ile Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Asn Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Leu Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ser Glu Asp Glu Ala Asp Tyr Tyr Cys His Ser Tyr Asp Gly Ser
85 90 95
Leu Ser Glu Gly Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 235
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 235
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Ile Ile Tyr Gly Asp Thr Lys Arg Ala Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Gly Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Asn Ser
85 90 95
Leu Ser Ala His Ala Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 236
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 236
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Gly Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Leu Phe Gly Asn Thr Asn Arg Pro Ser Gly Val Pro Gly Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Ser Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ser Asp Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Gly Gly Ser Val Ile Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 237
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 237
Gln Leu Val Leu Thr Gln Pro Ser Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Leu Gly Ala Pro
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Val Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Ser Ser Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 238
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 238
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Gly Val Thr Ile Ser Cys Thr Gly Asp Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Phe Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Glu Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Arg
85 90 95
Leu Gly Ala Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 239
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 239
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Lys Val Thr Phe Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln His Leu Pro Gly Ala Ala Pro Arg Leu
35 40 45
Leu Ile Tyr Ala Asn Thr Asn Arg Pro Leu Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Asn Arg
85 90 95
Leu Ser Gly Ser Leu Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 240
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 240
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Ala Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asp Ile Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Ile Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Gly Gly Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 241
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 241
Gln Phe Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Ser Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Asn Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Asn Gly Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 242
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 242
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Gly Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Leu Phe Gly Asn Thr Asn Arg Pro Ser Gly Val Pro Gly Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Ser Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ser Asp Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Gly Ser Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 243
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 243
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Gly Gly Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 244
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 244
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Thr Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Phe Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Asn Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Thr Asp Asp Glu Ala Asp Tyr Phe Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Arg Val Ser Ser Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 245
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 245
Gln Leu Val Leu Thr Gln Pro Ser Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Gly Ser Gly Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 246
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 246
Gln Phe Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Arg Ser Asn Phe Gly Ala Gly
20 25 30
His Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Asn Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ala Gly Leu
65 70 75 80
Gln Ser Asp Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Gly Ser Val Phe Gly Thr Gly Thr Lys Val Thr Val Leu
100 105 110
<210> 247
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 247
Gln Leu Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Val Gly Ala Gly
20 25 30
Tyr Asp Val His Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Asn Asn Arg Pro Ser Gly Val Pro Ala Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Arg
85 90 95
Leu Gly Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 248
<211> 232
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 248
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
20 25 30
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
35 40 45
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
50 55 60
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
65 70 75 80
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
85 90 95
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
100 105 110
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
115 120 125
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
130 135 140
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
145 150 155 160
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
165 170 175
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
180 185 190
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
195 200 205
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
210 215 220
Ser Leu Ser Leu Ser Pro Gly Lys
225 230
<210> 249
<211> 233
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 249
Glu Pro Arg Gly Pro Thr Ile Lys Pro Cys Pro Pro Cys Lys Cys Pro
1 5 10 15
Ala Pro Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys
20 25 30
Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val Thr Cys Val
35 40 45
Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser Trp Phe
50 55 60
Val Asn Asn Val Glu Val His Thr Ala Gln Thr Gln Thr His Arg Glu
65 70 75 80
Asp Tyr Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile Gln His
85 90 95
Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys
100 105 110
Asp Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly Ser
115 120 125
Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu Glu Met
130 135 140
Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe Met Pro
145 150 155 160
Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu Leu Asn
165 170 175
Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr Phe Met
180 185 190
Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg Asn Ser
195 200 205
Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His His Thr Thr
210 215 220
Lys Ser Phe Ser Arg Thr Pro Gly Lys
225 230
<210> 250
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 250
ccaggatggt tcttagactc ccc 23
<210> 251
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Artificial Sequence
<400> 251
caccagggtt tggaactggc 20
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| АНТИТЕЛА ПРОТИВ БЕЛКА-1 ЗАПРОГРАММИРОВАННОЙ КЛЕТОЧНОЙ СМЕРТИ (PD-1) И ИХ ПРИМЕНЕНИЕ | 2017 |
|
RU2725950C1 |
| АНТИТЕЛА ПРОТИВ VISTA И ИХ ПРИМЕНЕНИЕ | 2018 |
|
RU2750723C1 |
| БИСПЕЦИФИЧЕСКОЕ АНТИТЕЛО АНТИ-EGFR/АНТИ-4-1ВВ И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2831836C2 |
| АНТИТЕЛО ПРОТИВ ANG2 И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2788088C1 |
| Композиция для предупреждения и лечения заболевания кожи, содержащая вещество, специфично связывающееся с пептидом, имеющим происхождение из виментина | 2018 |
|
RU2751486C1 |
| АНТИТЕЛА К C10ORF54 И ИХ ПРИМЕНЕНИЯ | 2015 |
|
RU2819627C1 |
| АНТИТЕЛО ПРОТИВ TIE-2 И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2795455C2 |
| Биспецифическое антитело анти-HER2/анти-4-1BB и его применение | 2020 |
|
RU2831838C2 |
| ГУМАНИЗИРОВАННЫЕ АНТИТЕЛА И АНТИТЕЛА С СОЗРЕВШЕЙ АФФИННОСТЬЮ ПРОТИВ FcRH5 И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2748943C2 |
| СООТВЕТСТВУЮЩАЯ МИШЕНЬ ДЛЯ ЛЕЧЕНИЯ ФИБРОЗИРУЮЩИХ ЗАБОЛЕВАНИЙ И ЕЕ ПРИМЕНЕНИЕ | 2021 |
|
RU2830698C1 |

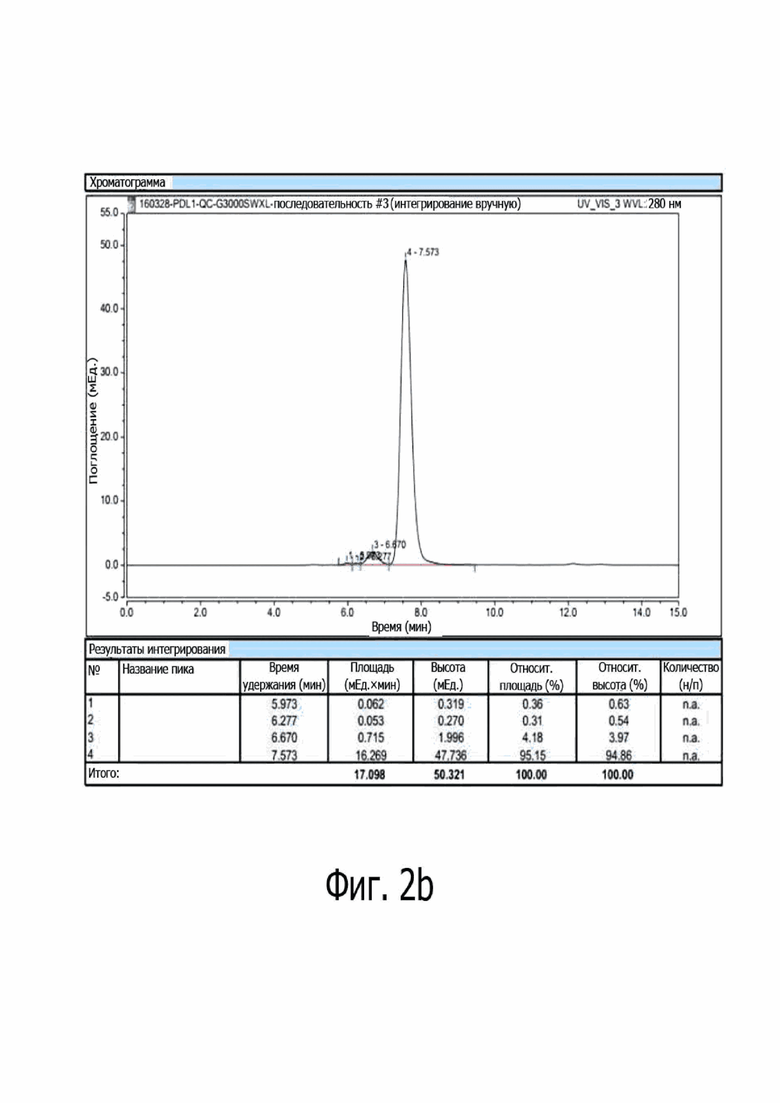
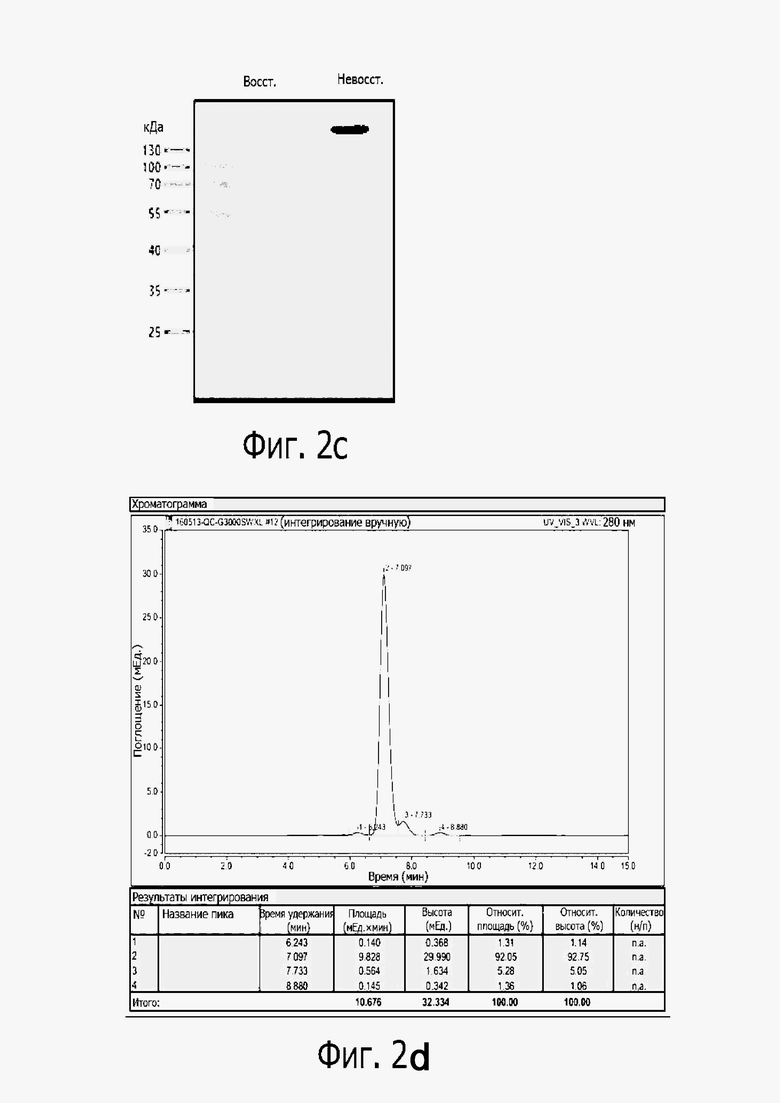
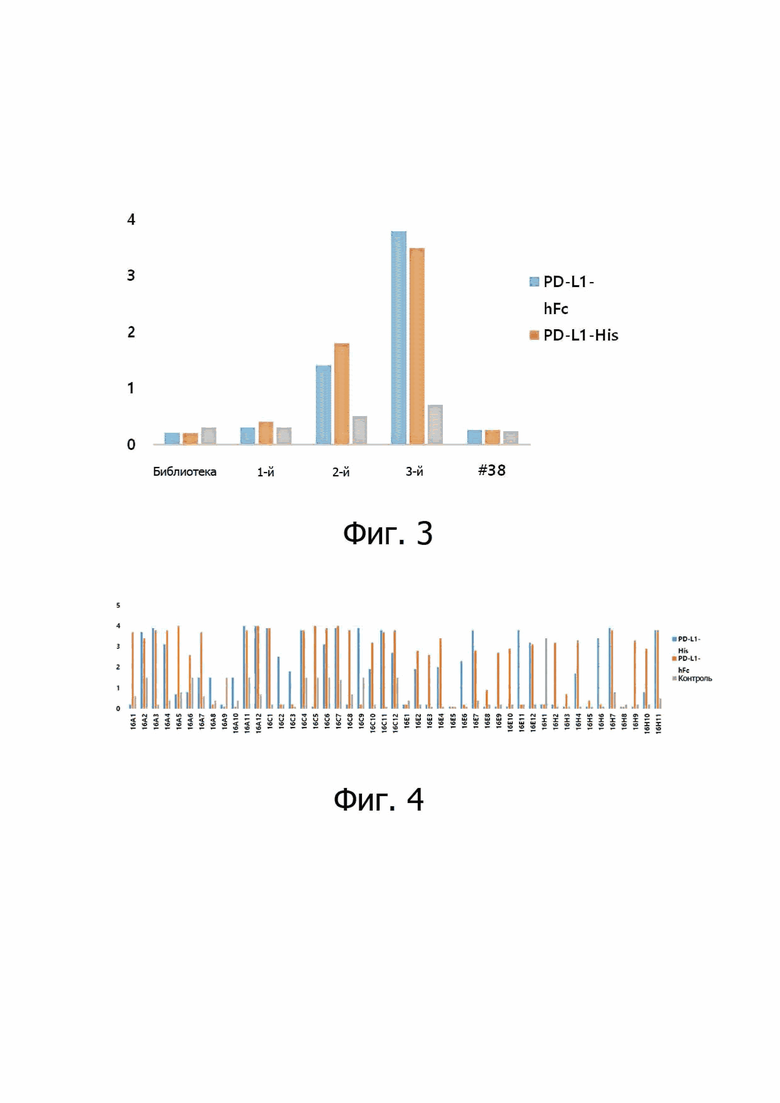
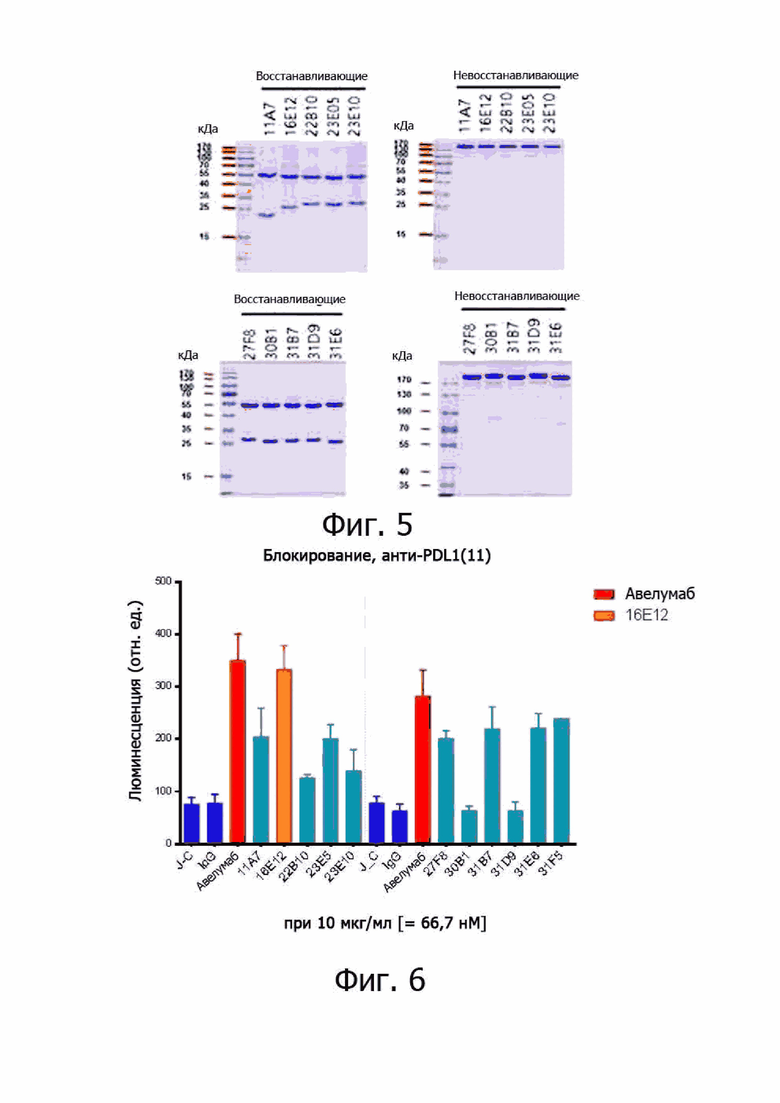
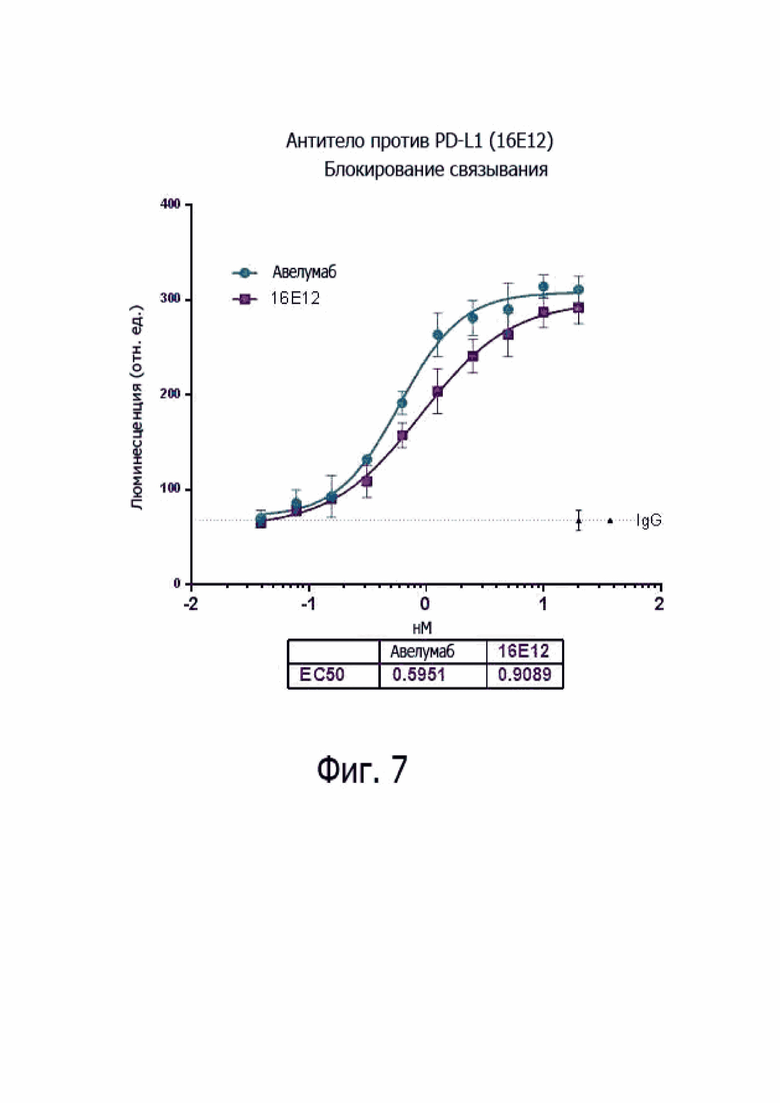
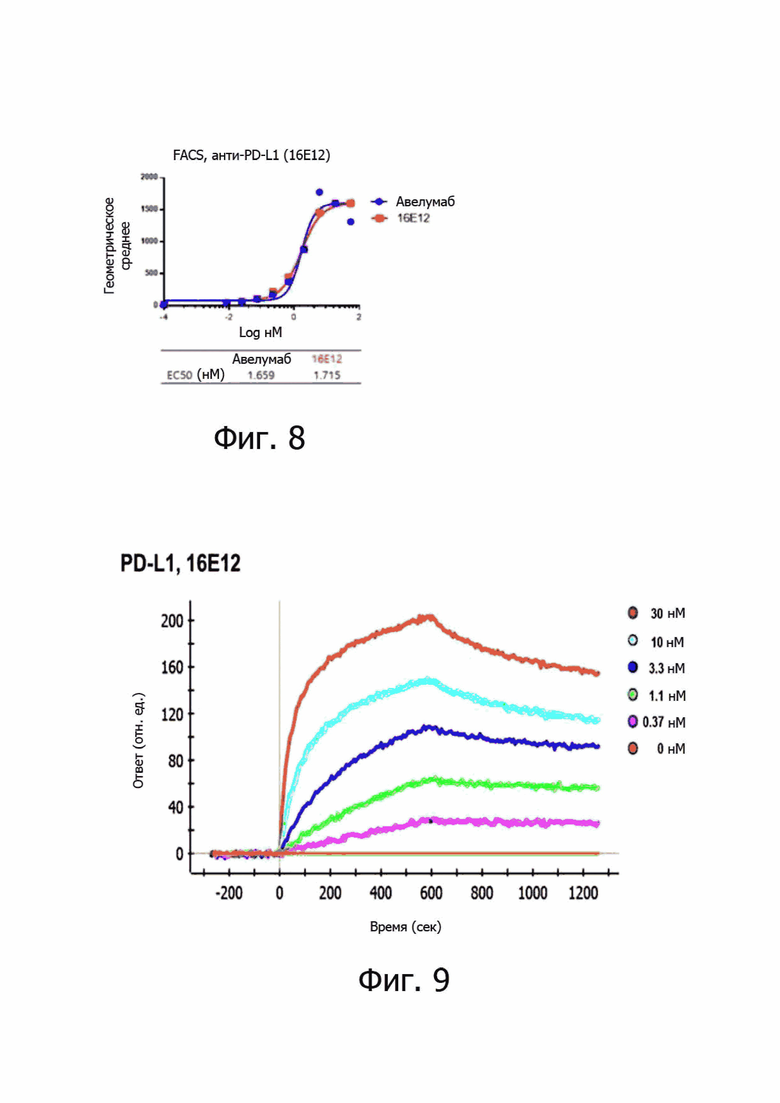
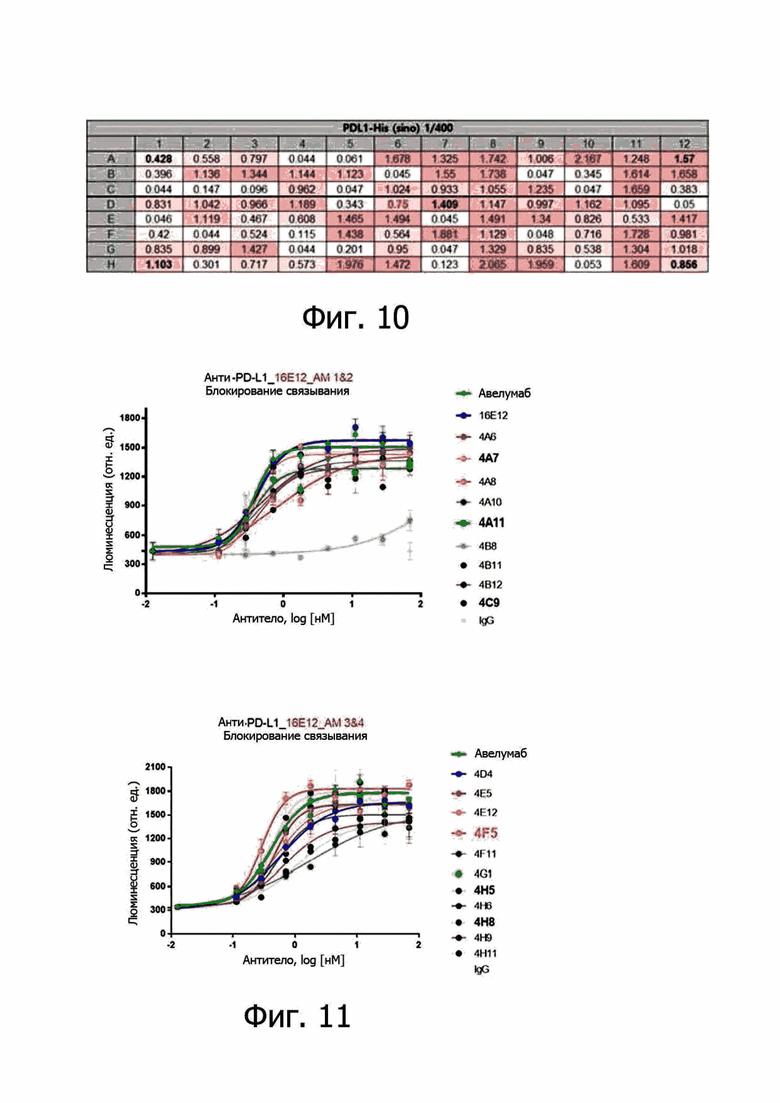
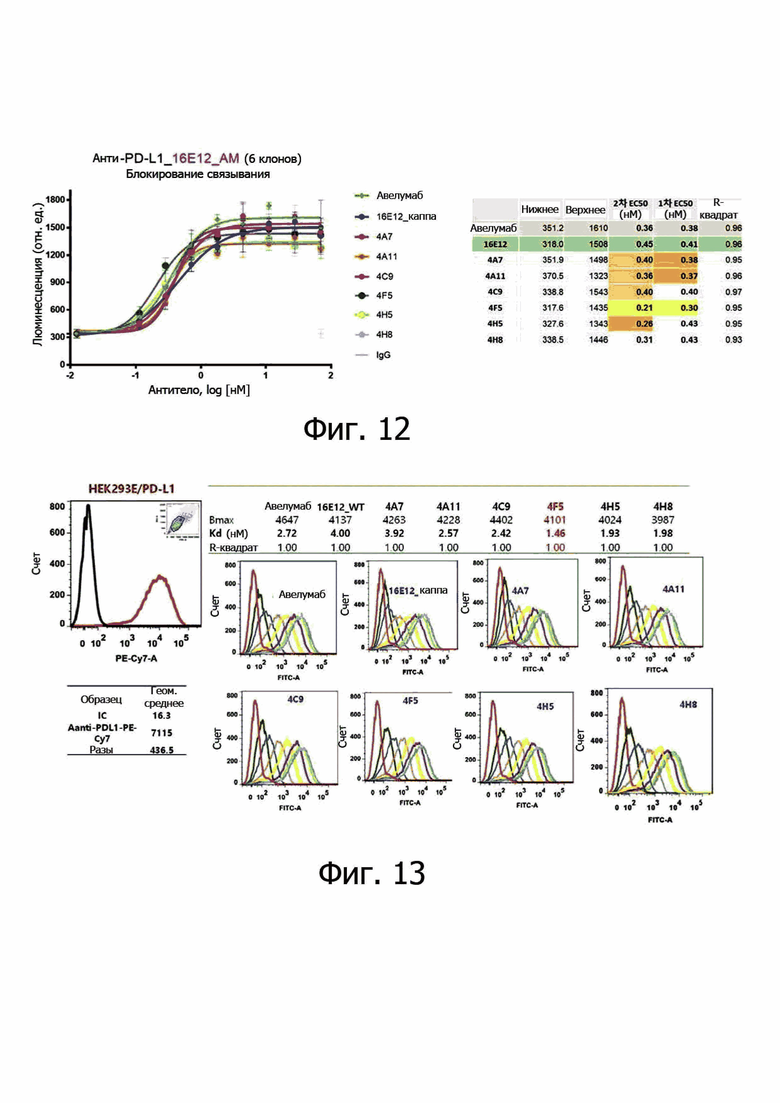
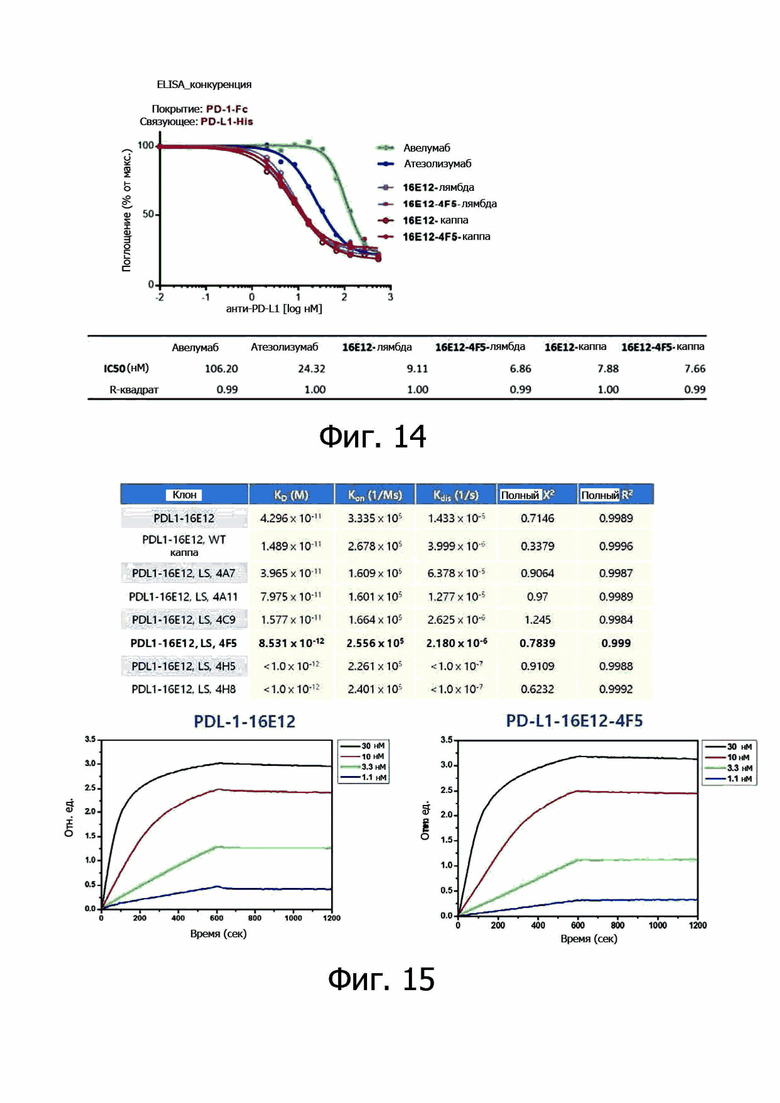
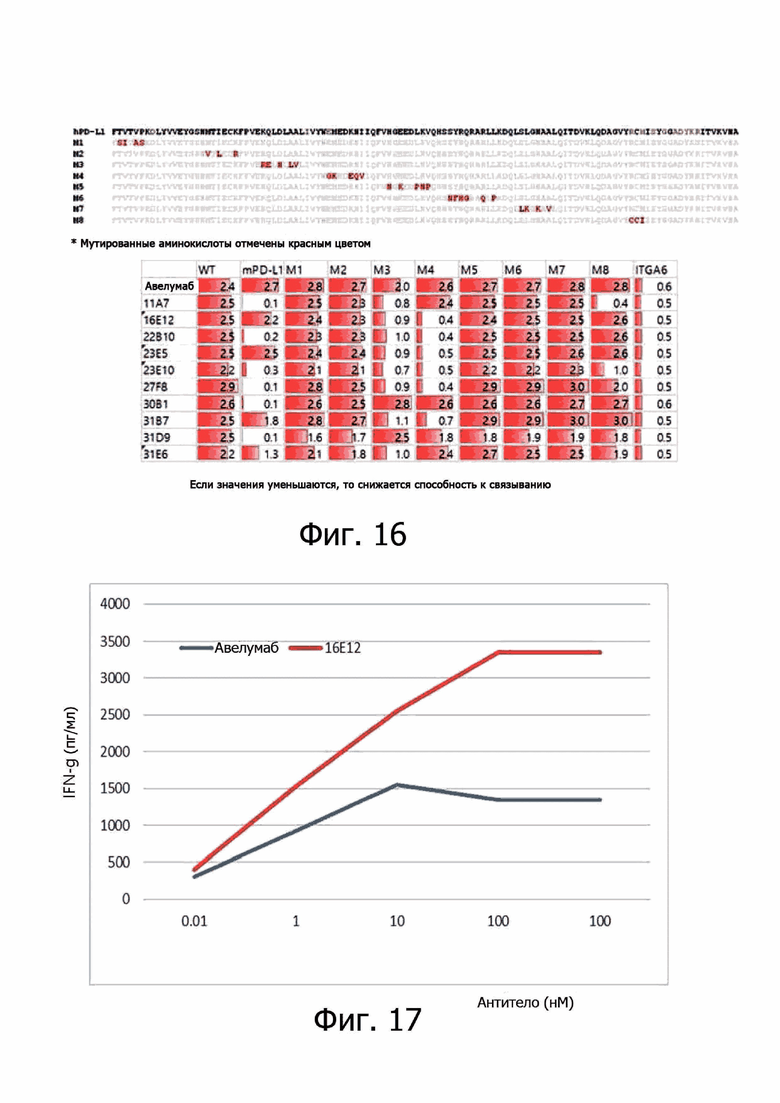
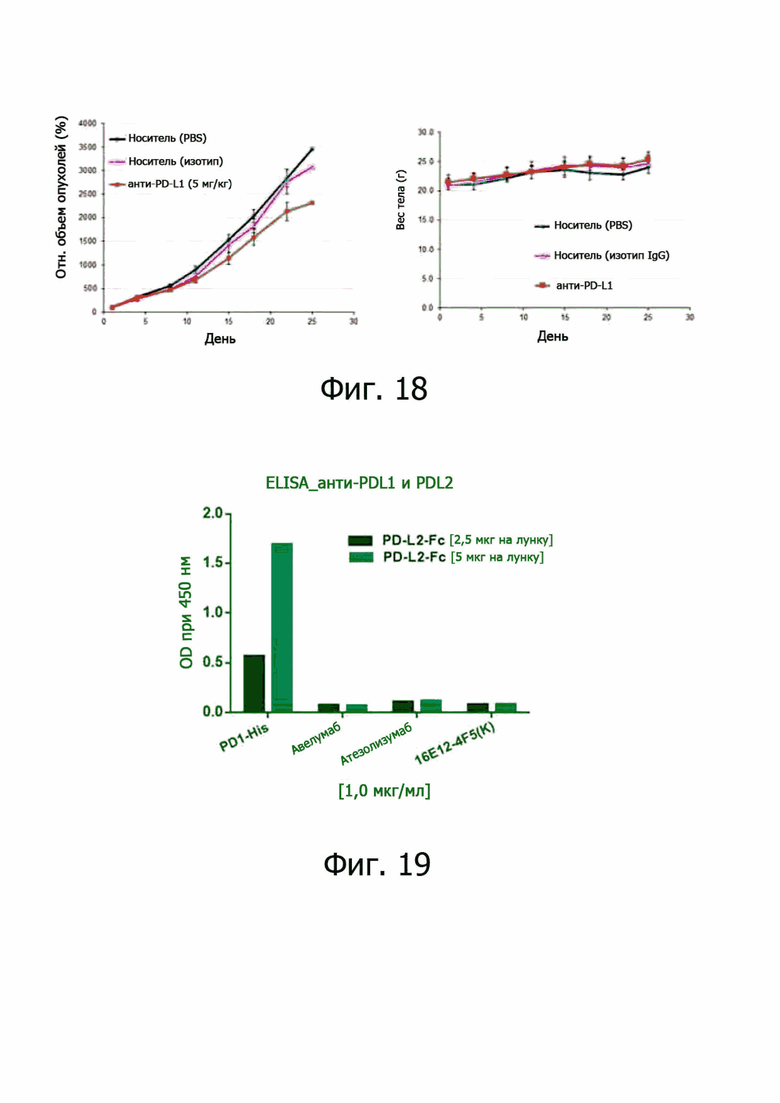
Группа изобретений относится к медицине, а именно к иммунологии и онкологии, и предназначена для профилактики или лечения рака либо инфекционных заболеваний. Представлены антитело против лиганда-1 запрограммированной клеточной смерти (PD-L1) человека либо его антигенсвязывающий фрагмент, кодирующая его нуклеиновая кислота, вектор, содержащий эту нуклеиновую кислоту, клетка, трансформированная этим вектором, способ получения данного антитела либо его антигенсвязывающего фрагмента и содержащая его композиция для профилактики или лечения рака либо инфекционных заболеваний. Группа изобретений позволяет улучшить активность и действие ингибированных опухолеспецифичных клеток Т-лимфоцитов путем ингибирования их функций с помощью моноклональных антител против PD-1 или его лиганда PD-L1 с тем, чтобы получить противораковые эффекты. 7 н. и 4 з.п. ф-лы, 13 табл., 12 пр., 19 ил.
1. Антитело, связывающееся с PD-L1, либо его антигенсвязывающий фрагмент, включающее:
вариабельную область тяжелой цепи, включающую:
CDR1 тяжелой цепи, последовательности SEQ ID NO: 2;
CDR2 тяжелой цепи, последовательности SEQ ID NO: 9; и
CDR3 тяжелой цепи, последовательности SEQ ID NO: 17; а также
вариабельную область легкой цепи, включающую:
CDR1 легкой цепи, последовательности SEQ ID NO: 89;
CDR2 легкой цепи, последовательности SEQ ID NO: 104; и
CDR3 легкой цепи, последовательности SEQ ID NO: 121, или
вариабельную область тяжелой цепи, включающую:
CDR1 тяжелой цепи, последовательности SEQ ID NO: 2;
CDR2 тяжелой цепи, последовательности SEQ ID NO: 9; и
CDR3 тяжелой цепи, последовательности SEQ ID NO: 17; а также
вариабельную область легкой цепи, включающую:
CDR1 легкой цепи, последовательности SEQ ID NO: 89;
CDR2 легкой цепи, последовательности SEQ ID NO: 119; и
CDR3 легкой цепи, последовательности SEQ ID NO: 139.
2. Антитело либо его антигенсвязывающий фрагмент по п. 1, включающее:
FR1 вариабельной области тяжелой цепи, выбранный из группы, состоящей из SEQ ID NOs: 26-34;
FR2 вариабельной области тяжелой цепи, выбранный из группы, состоящей из SEQ ID NOs: 35-41;
FR3 вариабельной области тяжелой цепи, выбранный из группы, состоящей из SEQ ID NOs: 42-49; и
FR4 вариабельной области тяжелой цепи, выбранный из группы, состоящей из SEQ ID NOs: 50-54.
3. Антитело либо его антигенсвязывающий фрагмент по п. 1, включающее:
FR1 вариабельной области легкой цепи, выбранный из группы, состоящей из SEQ ID NOs: 145-163;
FR2 вариабельной области легкой цепи, выбранный из группы, состоящей из SEQ ID NOs: 164-184;
FR3 вариабельной области легкой цепи, выбранный из группы, состоящей из SEQ ID NOs: 185-210; и
FR4 вариабельной области легкой цепи, выбранный из группы, состоящей из SEQ ID NOs: 211-216.
4. Антитело либо его антигенсвязывающий фрагмент по п. 1, включающее:
вариабельную область тяжелой цепи, включающую последовательность, приведенную в SEQ ID NO: 58 или 80.
5. Антитело либо его антигенсвязывающий фрагмент по п. 1, включающее:
вариабельную область легкой цепи, включающую последовательность, приведенную в SEQ ID NO: 218 или 240.
6. Нуклеиновая кислота, кодирующая антитело либо его антигенсвязывающий фрагмент по любому из пп. 1-5.
7. Экспрессионный вектор, содержащий нуклеиновую кислоту по п. 6.
8. Клетка для продуцирования антитела или его антигенсвязывающего фрагмента по любому из пп.1-5, которая трансформирована экспрессионным вектором по п. 7.
9. Способ получения антитела, связывающегося с PD-L1, либо его антигенсвязывающего фрагмента, включающий:
(a) культивирование клетки по п. 8; и
(b) извлечение антитела либо его антигенсвязывающего фрагмента из супернатантов клеточной культуры.
10. Композиция для профилактики или лечения рака, содержащая в качестве активного ингредиента антитело либо его антигенсвязывающий фрагмент по любому из пп. 1-5.
11. Композиция для профилактики или лечения инфекционных заболеваний, содержащая в качестве активного ингредиента антитело либо его антигенсвязывающий фрагмент по любому из пп. 1-5.
| US 20110209230 A1, 25.08.2011 | |||
| WO 2016090312 A1, 09.06.2016 | |||
| WO 2015097536 A2, 02.07.2015 | |||
| WO 2016061142 A1, 21.04.2016 | |||
| JÄGER V | |||
| et al | |||
| ПРИСПОСОБЛЕНИЕ ДЛЯ ПОДАЧИ УГЛЯ В ТЕНДЕР ПАРОВОЗА | 1920 |
|
SU293A1 |
| Насос | 1917 |
|
SU13A1 |
| KUHN P | |||
| et al | |||
| Recombinant antibodies for diagnostics and | |||
Авторы
Даты
2020-05-20—Публикация
2017-08-07—Подача