Родственные заявки и включение при помощи ссылки
Данная заявка заявляет приоритет предварительной заявки на патент США 61/836127, озаглавленной "Конструирование систем, способы и оптимизированные композиции для манипуляции с последовательностями" (Engineering Of Systems, Methods And Optimized Compositions For Sequence Manipulation), поданной 17 июня 2013 г. Данная заявка также заявляет приоритет предварительных заявок на патент США 61/758468; 61/769046; 61/802174; 61/806375; 61/814263; 61/819803 и 61/828130, каждая из которых озаглавлена "КОНСТРУИРОВАНИЕ И ОПТИМИЗАЦИЯ СИСТЕМ, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ" (ENGINEERING AND OPTIMIZATION OF SYSTEMS, METHODS AND COMPOSITIONS FOR SEQUENCE MANIPULATION), поданных 30 января 2013 г.; 25 февраля 2013 г.; 15 марта 2013 г.; 28 марта 2013 г.; 20 апреля 2013 г.; 6 мая 2013 г. и 28 мая 2013 г., соответственно. Также заявляется приоритет предварительных заявок на патент США 61/736527 и 61/748427, обе из которых озаглавлены "СИСТЕМЫ, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ" (SYSTEMS METHODS AND COMPOSITIONS FOR SEQUENCE MANIPULATION), поданных 12 декабря 2012 г. и 2 января 2013 г., соответственно. Также заявляется приоритет предварительных заявок на патент США 61/791409 и 61/835931, обе из которых озаглавлены BI-2011/008/44790.02.2003 и BI-2011/008/44790.03.2003, поданных 15 марта 2013 г. и 17 июня 2013 г., соответственно.
Также делается ссылка на предварительные заявки на патент США 61/835936, 61/836101, 61/836080, 61/836123 и 61/835973, каждая из которых подана 17 июня 2013 г.
Вышеприведенные заявки и все документы, цитируемые в них или во время их рассмотрения (“цитируемые документы заявки”), и все документы, цитируемые или упомянутые в цитируемых документах заявки, и все документы, цитируемые или упомянутые в данном документе (“документы, цитируемые в данном документе”), и все документы, цитируемые или упомянутые в документах, цитируемых в данном документе, совместно с любыми инструкциями изготовителя, описаниями, характеристиками продукта и технологическими картами для любых продуктов, упомянутых в данном документе или в любом документе, включенном с помощью ссылки в данный документ, таким образом, включены в данный документ с помощью ссылки и могут быть использованы в практическом осуществлении настоящего изобретения. Более конкретно, все документы, на которые ссылаются, включены при помощи ссылки в такой же мере, как если бы конкретно и отдельно было указано, что каждый отдельный документ включен при помощи ссылки.
Область техники
Настоящее изобретение в целом относится к системам, способам и композициям, применяемым для контроля экспрессии генов, включающего целенаправленное воздействие на последовательность, такое как внесение изменений в геном или редактирование гена, при котором можно использовать векторные системы, близкие к коротким палиндромным повторам, регулярно расположенным группами, (CRISPR) и их компонентам.
Утверждение касательно финансируемого из федерального бюджета исследования
Настоящее изобретение было разработано при правительственной поддержке, выданной Национальными институтами здравоохранения, NIH Pioneer Award DP1MH100706. Правительство обладает определенными правами на настоящее изобретение.
Предпосылки изобретения
Недавние достижения в технологиях секвенирования генома и способах анализа значительно ускорили возможность каталогизации и картирования генетических факторов, ассоциированных с широким разнообразием биологических функций и заболеваний. Технологии точного целенаправленного воздействия на геном необходимы для обеспечения систематичного обратного конструирования казуальных генетических изменений путем обеспечения возможности селективного внесения изменений в отдельные генетические элементы, а также для продвижения применений в области синтетической биологии, биотехнологии и медицины. Несмотря на то, что технологии редактирования генома, такие как конструктор доменов "цинковые пальцы", подобные транскрипционным активаторам эффекторы (TALE) или хоминг мегануклеазы, доступны для осуществления внесений изменений в целевой геном, все еще существует необходимость в новых технологиях конструирования генома, которые являются доступными, простыми в осуществлении, масштабируемыми и характеризуются возможностью целенаправленного воздействия на несколько положений в эукариотическом геноме.
Краткое описание изобретения
Существует актуальная необходимость в альтернативных и функциональных системах и технологиях для целенаправленного воздействия на последовательность с широким спектром применений. Настоящее изобретение удовлетворяет этой необходимости и предусматривает связанные с этим преимущества. CRISPR/Cas или система CRISPR-Cas (оба выражения используют взаимозаменяемо по всей данной заявке) не предусматривает получение индивидуализированных белков для целенаправленного воздействия на конкретные последовательности, но скорее один фермент Cas может быть запрограммирован короткой молекулой РНК для узнавания специфичной ДНК-мишени, другими словами, фермент Cas может связываться со специфичной ДНК-мишенью при помощи указанной короткой молекулы РНК. Добавление системы CRISPR-Cas к спектру технологий секвенирования генома и способов анализа может значительно упростить методику и ускорить возможность каталогизации и картирования генетических факторов, ассоциированных с широким разнообразием биологических функций и заболеваний. Для того чтобы эффективно использовать систему CRISPR-Cas для редактирования генома без вредного действия, важно понимать аспекты конструирования и оптимизации этих средств для конструирования генома, которые являются аспектами заявленного изобретения.
В одном аспекте настоящее изобретение предусматривает векторную систему, содержащую один или несколько векторов. В некоторых вариантах осуществления система содержит (a) первый регуляторный элемент, функционально связанный с парной tracr-последовательностью и одним или несколькими сайтами встраивания для встраивания одной или нескольких направляющих последовательностей выше парной tracr-последовательности, где при экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в клетке, к примеру, эукариотической клетке, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью; и (b) второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей указанный фермент CRISPR, содержащий последовательность ядерной локализации; где компоненты (a) и (b) находятся в одном и том же или в разных векторах системы. В некоторых вариантах осуществления компонент (a) дополнительно содержит tracr-последовательность ниже парной tracr-последовательности под контролем первого регуляторного элемента. В некоторых вариантах осуществления компонент (a) дополнительно содержит две или более направляющие последовательности, функционально связанные с первым регуляторным элементом, где при экспрессии каждая из двух или более направляющих последовательностей управляет специфичным к последовательности связыванием комплекса CRISPR со своей целевой последовательностью в эукариотической клетке. В некоторых вариантах осуществления система содержит tracr-последовательность под контролем третьего регуляторного элемента, такого как промотор полимеразы III. В некоторых вариантах осуществления tracr-последовательность характеризуется по меньшей мере 50%, 60%, 70%, 80%, 90%, 95% или 99% комплементарности последовательности по длине парной tracr-последовательности при оптимальном выравнивании. В некоторых вариантах осуществления комплекс CRISPR содержит одну или несколько последовательностей ядерной локализации, достаточно эффективных, чтобы управлять накоплением указанного комплекса CRISPR в обнаруживаемом количестве в ядре эукариотической клетки. Не желая быть связанными теорией, полагают, что последовательность ядерной локализации не является необходимой для активности комплекса CRISPR у эукариот, но что включение таких последовательностей повышает активность системы, особенно в отношении нацеливания на молекулы нуклеиновых кислот в ядре. В некоторых вариантах осуществления фермент CRISPR является ферментом системы CRISPR II типа. В некоторых вариантах осуществления фермент CRISPR является ферментом Cas9. В некоторых вариантах осуществления фермент Cas9 представляет собой Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. Фермент может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления фермент CRISPR кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В некоторых вариантах осуществления у фермента CRISPR отсутствует активность для расщепления нитей ДНК. В некоторых вариантах осуществления первый регуляторный элемент является промотором полимеразы III. В некоторых вариантах осуществления второй регуляторный элемент является промотором полимеразы II. В некоторых вариантах осуществления направляющая последовательность составляет по меньшей мере 15, 16, 17, 18, 19, 20, 25 нуклеотидов, или от 10 до 30, или от 15 до 25, или от 15 до 20 нуклеотидов в длину. В целом и по всему данному описанию выражение “вектор” относится к молекуле нуклеиновой кислоты, способной переносить другую нуклеиновую кислоту, с которой она была связана. Векторы включают, без ограничения, молекулы нуклеиновых кислот, которые являются одноцепочечными, двухцепочечными или частично двухцепочечными; молекулы нуклеиновых кислот, которые содержат один или несколько свободных концов, не содержат свободных концов (к примеру, кольцевые); молекулы нуклеиновых кислот, которые содержат ДНК, РНК или и ту, и другую; и другие разновидности полинуклеотидов, известных в уровне техники. Одним типом вектора является “плазмида”, которая означает кольцевую петлю двухцепочечной ДНК, в которую можно встраивать дополнительные сегменты ДНК, как, например, при помощи стандартных технологий молекулярного клонирования. Другим типом вектора является вирусный вектор, где полученные из вируса последовательности ДНК или РНК присутствуют в векторе для упаковки в вирус (к примеру, ретровирусы, ретровирусы с дефективной системой репликации, аденовирусы, аденовирусы с дефективной системой репликации и аденоассоциированные вирусы). Вирусные векторы также включают полинуклеотиды, переносимые вирусами для трансфекции клетки-хозяина. Определенные векторы способны к саморегулируемой репликации в клетке-хозяине, в которую они введены (к примеру, бактериальные векторы с бактериальной точкой начала репликации и эписомные векторы для млекопитающих). Другие векторы (к примеру, векторы для млекопитающих, отличные от эписомных) интегрируются в геном клетки-хозяина после введения в клетку-хозяина и, таким образом, реплицируются наряду с геномом хозяина. Более того, определенные векторы способны управлять экспрессией генов, с которыми они функционально связаны. Такие векторы в данном документе называют “векторами экспрессии”. Общепринятые пригодные в технологиях рекомбинантной ДНК векторы экспрессии часто находятся в форме плазмид.
Рекомбинантные векторы экспрессии могут содержать нуклеиновую кислоту согласно настоящему изобретению в форме, подходящей для экспрессии нуклеиновой кислоты в клетке-хозяине, что означает, что рекомбинантные векторы экспрессии включают один или несколько регуляторных элементов, которые могут быть выбраны с учетом клеток-хозяев, которые предполагается использовать для экспрессии, которые функционально связаны с последовательностью нуклеиновой кислоты, экспрессия которой предполагается. В контексте рекомбинантного вектора экспрессии выражение “функционально связанный” предназначено означать, что представляющая интерес нуклеотидная последовательность связана с регуляторным(и) элементом(ами) таким образом, при котором обеспечивается экспрессия нуклеотидной последовательности (к примеру, в in vitro системе транскрипции/трансляции или в клетке-хозяине, когда вектор вводят в клетку-хозяина).
Выражение “регуляторный элемент” предназначено включать промоторы, энхансеры, участки внутренней посадки рибосомы (IRES) и другие контролирующие экспрессию элементы (к примеру, сигналы терминации транскрипции, такие как сигналы полиаденилирования и поли-U-последовательности). Такие регуляторные элементы описаны, например, в Goeddel, GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990). Регуляторные элементы включают такие, которые управляют конститутивной экспрессией нуклеотидной последовательности во многих типах клеток-хозяев, и такие, которые управляют экспрессией нуклеотидной последовательности только в определенных клетках-хозяевах (к примеру, тканеспецифичные регуляторные последовательности). Тканеспецифичный промотор может управлять экспрессией преимущественно в представляющей интерес целевой ткани, такой как мышца, нейрон, кость, кожа, кровь, конкретных органах (к примеру, печени, поджелудочной железе) или определенных типах клеток (к примеру, лимфоцитах). Регуляторные элементы также могут управлять экспрессией зависимым от времени образом, как, например, зависимым от клеточного цикла или зависимым от стадии развития образом, который может быть или может не быть также тканеспецифичным или специфичным к типу клеток. В некоторых вариантах осуществления вектор содержит один или несколько промоторов pol III (к примеру, 1, 2, 3, 4, 5 или более промоторов pol III), один или несколько промоторов pol II (к примеру, 1, 2, 3, 4, 5 или более промоторов pol II), один или несколько промоторов pol I (к примеру, 1, 2, 3, 4, 5 или более промоторов pol I) или их комбинации. Примеры промоторов pol III включают без ограничения промоторы U6 и H1. Примеры промоторов pol II включают без ограничения ретровирусный промотор LTR вируса саркомы Рауса (RSV) (необязательно с энхансером RSV), промотор цитомегаловируса (CMV) (необязательно с энхансером CMV) [см., например, Boshart et al, Cell, 41:521-530 (1985)], промотор SV40, промотор дигидрофолатредуктазы, промотор β-актина, промотор глицерофосыаткиназы (PGK) и промотор EF1α. Также выражением “регуляторный элемент” охвачены энхансерные элементы, такие как WPRE; энхансеры CMV; сегмент R-U5' в LTR HTLV-I (Mol. Cell. Biol., Vol. 8(1), p. 466-472, 1988); энхансер SV40; и интронная последовательность между экзонами 2 и 3 β-глобина кролика (Proc. Natl. Acad. Sci. USA., Vol. 78(3), p. 1527-31, 1981). Специалистам в данной области будет понятно, что структура вектора экспрессии может зависеть от таких факторов, как выбор клетки хозяина, подлежащей трансформации, желательный уровень экспрессии и т. п. Вектор можно вводить в клетки-хозяева с получением, таким образом, транскриптов, белков или пептидов, в том числе слитых белков или пептидов, кодируемых нуклеиновыми кислотами, которые описаны в данном документе (к примеру, транскриптов коротких палиндромных повторов, регулярно расположенных группами (CRISPR), белков, ферментов, их мутантных форм, их слитых белков и т.п.).
Преимущественные векторы включают лентивирусы и аденоассоциированные вирусы, и типы таких векторов также могут быть выбраны для целенаправленного воздействия на определенные типы клеток.
В одном аспекте настоящее изобретение предусматривает вектор, содержащий регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, содержащий одну или несколько последовательностей ядерной локализации. В некоторых вариантах осуществления указанный регуляторный элемент управляет транскрипцией фермента CRISPR в эукариотической клетке, так что указанный фермент CRISPR накапливается в обнаруживаемом количестве в ядре эукариотической клетки. В некоторых вариантах осуществления регуляторный элемент является промотором полимеразы II. В некоторых вариантах осуществления фермент CRISPR является ферментом системы CRISPR II типа. В некоторых вариантах осуществления фермент CRISPR является ферментом Cas9. В некоторых вариантах осуществления фермент Cas9 представляет собой Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. В некоторых вариантах осуществления фермент CRISPR кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В некоторых вариантах осуществления у фермента CRISPR отсутствует активность для расщепления нитей ДНК.
В одном аспекте настоящее изобретение предусматривает фермент CRISPR, содержащий одну или несколько последовательностей ядерной локализации, достаточно эффективных, чтобы управлять накоплением указанного фермента CRISPR в обнаруживаемом количестве в ядре эукариотической клетки. В некоторых вариантах осуществления фермент CRISPR является ферментом системы CRISPR II типа. В некоторых вариантах осуществления фермент CRISPR является ферментом Cas9. В некоторых вариантах осуществления фермент Cas9 представляет собой Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. Фермент может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления у фермента CRISPR отсутствует способность расщеплять одну или несколько нитей целевой последовательности, с которой он связывается.
В одном аспекте настоящее изобретение предусматривает эукариотическую клетку-хозяина, содержащую (a) первый регуляторный элемент, функционально связанный с парной tracr-последовательностью и одним или несколькими сайтами встраивания для встраивания одной или нескольких направляющих последовательностей выше парной tracr-последовательности, где при экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в эукариотической клетке, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью; и/или (b) второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей указанный фермент CRISPR, содержащий последовательность ядерной локализации. В некоторых вариантах осуществления клетка-хозяин содержит компоненты (a) и (b). В некоторых вариантах осуществления компонент (a), компонент (b) или компоненты (a) и (b) стабильно интегрируются в геном эукариотической клетки-хозяина. В некоторых вариантах осуществления компонент (a) дополнительно содержит tracr-последовательность ниже парной tracr-последовательности под контролем первого регуляторного элемента. В некоторых вариантах осуществления компонент (a) дополнительно содержит две или более направляющие последовательности, функционально связанные с первым регуляторным элементом, где при экспрессии каждая из двух или более направляющих последовательностей управляет специфичным к последовательности связыванием комплекса CRISPR со своей целевой последовательностью в эукариотической клетке. В некоторых вариантах осуществления эукариотическая клетка-хозяин дополнительно содержит третий регуляторный элемент, такой как промотор полимеразы III, функционально связанный с указанной tracr-последовательностью. В некоторых вариантах осуществления tracr-последовательность характеризуется по меньшей мере 50%, 60%, 70%, 80%, 90%, 95% или 99% комплементарности последовательности по длине парной tracr-последовательности при оптимальном выравнивании. В некоторых вариантах осуществления фермент CRISPR содержит одну или несколько последовательностей ядерной локализации, достаточно эффективных, чтобы управлять накоплением указанного фермента CRISPR в обнаруживаемом количестве в ядре эукариотической клетки. В некоторых вариантах осуществления фермент CRISPR является ферментом системы CRISPR II типа. В некоторых вариантах осуществления фермент CRISPR является ферментом Cas9. В некоторых вариантах осуществления фермент Cas9 представляет собой Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. Фермент может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления фермент CRISPR кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В некоторых вариантах осуществления у фермента CRISPR отсутствует активность для расщепления нитей ДНК. В некоторых вариантах осуществления первый регуляторный элемент является промотором полимеразы III. В некоторых вариантах осуществления второй регуляторный элемент является промотором полимеразы II. В некоторых вариантах осуществления направляющая последовательность составляет по меньшей мере 15, 16, 17, 18, 19, 20, 25 нуклеотидов, или от 10 до 30, или от 15 до 25, или от 15 до 20 нуклеотидов в длину. В одном аспекте настоящее изобретение предусматривает отличный от человека эукариотический организм, предпочтительно многоклеточный эукариотический организм, содержащий эукариотическую клетку-хозяина согласно любому из описанных вариантов осуществления. В других аспектах настоящее изобретение предусматривает эукариотический организм, предпочтительно многоклеточный эукариотический организм, содержащий эукариотическую клетку-хозяина согласно любому из описанных вариантов осуществления. Организм в некоторых вариантах осуществления данных аспектов может быть животным, например, млекопитающим. Также организмом может быть членистоногое, как, например, насекомое. Организмом также может быть растение. Кроме того, организмом может быть гриб.
В одном аспекте настоящее изобретение предусматривает набор, содержащий один или несколько компонентов, описанных в данном документе. В некоторых вариантах осуществления набор содержит векторную систему и инструкции по применению набора. В некоторых вариантах осуществления векторная система содержит (a) первый регуляторный элемент, функционально связанный с парной tracr-последовательностью и одним или несколькими сайтами встраивания для встраивания одной или нескольких направляющих последовательностей выше парной tracr-последовательности, где при экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в эукариотической клетке, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью; и/или (b) второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей указанный фермент CRISPR, содержащий последовательность ядерной локализации. В некоторых вариантах осуществления набор содержит компоненты (a) и (b), находящиеся в одном и том же или в разных векторах системы. В некоторых вариантах осуществления компонент (a) дополнительно содержит tracr-последовательность ниже парной tracr-последовательности под контролем первого регуляторного элемента. В некоторых вариантах осуществления компонент (a) дополнительно содержит две или более направляющие последовательности, функционально связанные с первым регуляторным элементом, где при экспрессии каждая из двух или более направляющих последовательностей управляет специфичным к последовательности связыванием комплекса CRISPR со своей целевой последовательностью в эукариотической клетке. В некоторых вариантах осуществления система дополнительно содержит третий регуляторный элемент, такой как промотор полимеразы III, функционально связанный с указанной tracr-последовательностью. В некоторых вариантах осуществления tracr-последовательность характеризуется по меньшей мере 50%, 60%, 70%, 80%, 90%, 95% или 99% комплементарности последовательности по длине парной tracr-последовательности при оптимальном выравнивании. В некоторых вариантах осуществления фермент CRISPR содержит одну или несколько последовательностей ядерной локализации, достаточно эффективных, чтобы управлять накоплением указанного фермента CRISPR в обнаруживаемом количестве в ядре эукариотической клетки. В некоторых вариантах осуществления фермент CRISPR является ферментом системы CRISPR II типа. В некоторых вариантах осуществления фермент CRISPR является ферментом Cas9. В некоторых вариантах осуществления фермент Cas9 представляет собой Cas9 S. pneumoniae, S. pyogenes или S. thermophilus и может включать мутированный Cas9, полученный из этих организмов. Фермент может быть гомологом или ортологом Cas9. В некоторых вариантах осуществления фермент CRISPR кодон-оптимизирован для экспрессии в эукариотической клетке. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или двух нитей в определенной точке целевой последовательности. В некоторых вариантах осуществления у фермента CRISPR отсутствует активность для расщепления нитей ДНК. В некоторых вариантах осуществления первый регуляторный элемент является промотором полимеразы III. В некоторых вариантах осуществления второй регуляторный элемент является промотором полимеразы II. В некоторых вариантах осуществления направляющая последовательность составляет по меньшей мере 15, 16, 17, 18, 19, 20, 25 нуклеотидов, или от 10 до 30, или от 15 до 25, или от 15 до 20 нуклеотидов в длину.
В одном аспекте настоящее изобретение предусматривает способ модификации целевого полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает обеспечение связывания комплекса CRISPR с целевым полинуклеотидом для осуществления расщепления указанного целевого полинуклеотида с модификацией, таким образом, целевого полинуклеотида, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизирующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизируется с tracr-последовательностью. В некоторых вариантах осуществления указанное расщепление включает расщепление одной или двух нитей в определенной точке целевой последовательности указанным ферментом CRISPR. В некоторых вариантах осуществления указанное расщепление приводит к сниженной транскрипции целевого гена. В некоторых вариантах осуществления способ дополнительно включает репарацию указанного расщепленного целевого полинуклеотида при помощи гомологичной рекомбинации с экзогенным матричным полинуклеотидом, где указанная репарация приводит к мутации, включающей вставку, делецию или замену одного или нескольких нуклеотидов указанного целевого полинуклеотида. В некоторых вариантах осуществления указанная мутация приводит к одной или нескольким аминокислотным заменам в белке, экспрессируемом с гена, содержащего целевую последовательность. В некоторых вариантах осуществления способ дополнительно включает доставку одного или нескольких векторов в указанную эукариотическую клетку, где один или несколько векторов управляют экспрессией одного или нескольких из: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности. В некоторых вариантах осуществления указанные векторы доставляют в эукариотическую клетку в субъекте. В некоторых вариантах осуществления указанная модификация имеет место в указанной эукариотической клетке в клеточной культуре. В некоторых вариантах осуществления способ дополнительно включает выделение указанной эукариотической клетки из субъекта перед указанной модификацией. В некоторых вариантах осуществления способ дополнительно включает возвращение указанной эукариотической клетки и/или клеток, полученных из субъекта, указанному субъекту.
В одном аспекте настоящее изобретение предусматривает способ модификации экспрессии полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает обеспечение связывания комплекса CRISPR с полинуклеотидом так, что указанное связывание приводит к повышенной или пониженной экспрессии указанного полинуклеотида; где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизирующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизируется с tracr-последовательностью. В некоторых вариантах осуществления способ дополнительно включает доставку одного или нескольких векторов в указанные эукариотические клетки, где один или несколько векторов управляют экспрессией одного или нескольких из: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности.
В одном аспекте настоящее изобретение предусматривает способ получения модельной эукариотической клетки, содержащей мутированный ген, ответственный за развитие заболевания. В некоторых вариантах осуществления ген, ответственный за развитие заболевания, является любым геном, ассоциированным с повышением риска наличия или развития заболевания. В некоторых вариантах осуществления способ включает (a) введение одного или нескольких векторов в эукариотическую клетку, где один или несколько векторов управляют экспрессией одного или нескольких из: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности; и (b) обеспечение связывания комплекса CRISPR с целевым полинуклеотидом для осуществления расщепления целевого полинуклеотида в указанном гене, ответственном за развитие заболевания, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью в целевом полинуклеотиде, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью, таким образом, получая модельную эукариотическую клетку, содержащую мутированный ген, ответственный за развитие заболевания. В некоторых вариантах осуществления указанное расщепление включает расщепление одной или двух нитей в определенной точке целевой последовательности указанным ферментом CRISPR. В некоторых вариантах осуществления указанное расщепление приводит к сниженной транскрипции целевого гена. В некоторых вариантах осуществления способ дополнительно включает репарацию указанного расщепленного целевого полинуклеотида при помощи гомологичной рекомбинации с экзогенным матричным полинуклеотидом, где указанная репарация приводит к мутации, включающей вставку, делецию или замену одного или нескольких нуклеотидов указанного целевого полинуклеотида. В некоторых вариантах осуществления указанная мутация приводит к одной или нескольким аминокислотным заменам при экспрессии белка с гена, содержащего целевую последовательность.
В одном аспекте настоящее изобретение предусматривает способ получения биологически активного средства, которое модулирует процесс передачи сигнала в клетке, ассоциированный с геном, ответственным за развитие заболевания. В некоторых вариантах осуществления ген, ответственный за развитие заболевания, является любым геном, ассоциированным с повышением риска наличия или развития заболевания. В некоторых вариантах осуществления способ включает (a) приведение тестового соединения в контакт с модельной клеткой по любому одному из описанных вариантов осуществления и (b) обнаружение изменения при считывании, которое свидетельствует об уменьшении или усилении процесса передачи сигнала в клетке, ассоциированного с указанной мутацией в указанном гене, ответственном за развитие заболевания, с получением, таким образом, указанного биологически активного средства, которое модулирует указанный процесс передачи сигнала в клетке, ассоциированный с указанным геном, ответственным за развитие заболевания.
В одном аспекте настоящее изобретение предусматривает рекомбинантный полинуклеотид, содержащий направляющую последовательность выше парной tracr-последовательности, где направляющая последовательность при экспрессии управляет специфичным к последовательности связыванием комплекса CRISPR с соответствующей целевой последовательностью, присутствующей в эукариотической клетке. В некоторых вариантах осуществления целевая последовательность является вирусной последовательностью, присутствующей в эукариотической клетке. В некоторых вариантах осуществления целевая последовательность является протоонкогеном или онкогеном.
В одном аспекте настоящее изобретение предусматривает способ отбора одной или нескольких прокариотических клеток путем введения одной или нескольких мутаций в ген в одной или нескольких прокариотических клетках, при этом способ включает введение одного или нескольких векторов в прокариотическую(ие) клетку(и), где один или несколько векторов управляют экспрессией одного или нескольких из: фермента CRISPR, направляющей последовательности, связанной с парной tracr-последовательностью, tracr-последовательности и матрицы редактирования; где матрица редактирования содержит одну или несколько мутаций, которые прекращают расщепление фермента CRISPR; обеспечение гомологичной рекомбинации матрицы редактирования с целевым полинуклеотидом в отбираемой(ых) клетке(ах); обеспечение связывания комплекса CRISPR с целевым полинуклеотидом для осуществления расщепления целевого полинуклеотида в указанном гене, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью в целевом полинуклеотиде, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью, где связывание комплекса CRISPR с целевым полинуклеотидом индуцирует гибель клеток, с обеспечением тем самым отбора одной или нескольких прокариотических клеток, в которые были введены одна или несколько мутаций. В предпочтительном варианте осуществления фермент CRISPR представляет собой Cas9. В другом аспекте настоящего изобретения отбираемая клетка может быть эукариотической клеткой. Аспекты настоящего изобретения предусматривают отбор конкретных клеток без необходимости наличия маркера отбора или двухстадийного способа, который может включать систему негативного отбора.
В некоторых аспектах настоящее изобретение предусматривает не встречающуюся в природе или сконструированную композицию, содержащую полинуклеотидную последовательность химерной РНК (chiRNA) системы CRISPR-CAS, где полинуклеотидная последовательность содержит (a) направляющую последовательность, способную гибридизироваться с целевой последовательностью в эукариотической клетке, (b) парную tracr-последовательность и (c) tracr-последовательность, где (a), (b) и (c) расположены в 5'-3' ориентации, где при транскрипции парная tracr-последовательность гибридизируется с tracr-последовательностью, а направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью,
или
ферментную систему CRISPR, где система кодируется векторной системой, содержащей один или несколько векторов, которые содержат I. первый регуляторный элемент, функционально связанный с полинуклеотидной последовательностью химерной РНК (chiRNA) системы CRISPR-CAS, где полинуклеотидная последовательность содержит (a) одну или несколько направляющих последовательностей, способных гибридизироваться с одной или несколькими целевыми последовательностями в эукариотической клетке, (b) парную tracr-последовательность и (c) одну или несколько tracr-последовательностей, и II. второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, содержащий по меньшей мере одну или несколько последовательностей ядерной локализации, где (a), (b) и (c) расположены в 5'-3' ориентации, где компоненты I и II находятся в одном и том же или в разных векторах системы, где при транскрипции парная tracr-последовательность гибридизируется с tracr-последовательностью, а направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью, или мультиплексную ферментную систему CRISPR, где система кодируется векторной системой, содержащей один или несколько векторов, которые содержат I. первый регуляторный элемент, функционально связанный с (a) одним или несколькими направляющими последовательностями, способными гибридизироваться с целевой последовательностью в клетке, и (b) по меньшей мере одной или несколькими парными tracr-последовательностями, II. второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, и III. третий регуляторный элемент, функционально связанный с tracr-последовательностью, где компоненты I, II и III находятся в одном и том же или в разных векторах системы, где при транскрипции парная tracr-последовательность гибридизируется с tracr-последовательностью, а направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью, и где в мультиплексной системе используется множество направляющих последовательностей и одна tracr-последовательность; и где одна или несколько из направляющих, tracr- и парных tracr-последовательностей модифицируются с повышением стабильности.
В аспектах настоящего изобретения модификация включает сконструированную вторичную структуру. Например, модификация может включать уменьшение участка гибридизации между парной tracr-последовательностью и tracr-последовательностью. Например, модификация также может включать слияние парной tracr-последовательности и tracr-последовательности посредством искусственной петли. Модификация может включать tracr-последовательность с длиной от 40 до 120 п.о. В вариантах осуществления настоящего изобретения tracr-последовательность составляет от 40 п.о. до полной длины tracr. В определенных вариантах осуществления длина tracRNA включает по меньшей мере нуклеотиды 1-67, а в некоторых вариантах осуществления по меньшей мере нуклеотиды 1-85 tracRNA дикого типа. В некоторых вариантах осуществления можно использовать по меньшей мере нуклеотиды, соответствующие нуклеотидам 1-67 или 1-85 tracRNA Cas9 S. pyogenes дикого типа. В тех случаях, когда в системе CRISPR используются ферменты, отличные от Cas9 или отличные от SpCas9, тогда в релевантной tracRNA дикого типа могут присутствовать соответствующие нуклеотиды. В некоторых вариантах осуществления длина tracRNA включает не более чем нуклеотиды 1-67 или 1-85 tracRNA дикого типа. Модификация может включать оптимизацию последовательности. В определенных аспектах оптимизация последовательности может включать снижение частоты встречаемости полиТ-последовательностей в tracr- и/или парной tracr-последовательности. Оптимизацию последовательности можно совмещать с уменьшением участка гибридизации между парной tracr-последовательностью и tracr-последовательностью; например, tracr-последовательностью уменьшенной длины.
В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает уменьшение полиТ-последовательностей в tracr- и/или парной tracr-последовательности. В некоторых аспектах настоящего изобретения один или несколько T, присутствующих в полиТ-последовательности соответствующей последовательности дикого типа (то есть, фрагмент из более 3, 4, 5, 6 или более смежных T-оснований; в некоторых вариантах осуществления фрагмент из не более 10, 9, 8, 7, 6 смежных T-оснований), могут быть заменены на отличный от T нуклеотид, к примеру, A, так что цепочка распадается на меньшие фрагменты из T, при этом каждый фрагмент имеет 4 или менее 4 (например, 3 или 2) смежных T. Основания, отличные от A можно использовать для замены, например, C или G, или не встречающиеся в природе нуклеотиды, или модифицированные нуклеотиды. Если цепочка из T участвует в образовании "шпильки" (или структуры по типу "петля-на-стебле"), тогда предпочтительно, чтобы комплементарное основание для отличного от T основания было изменено на комплементарное отличному от T нуклеотиду. Например, если отличным от T основанием является A, тогда его комплементарное основание может быть изменено на T, к примеру, для сохранения или содействия сохранению вторичной структуры. К примеру, 5'-TTTTT может быть изменено с получением 5'-TTTAT, а комплементарная 5'-AAAAA может быть изменена на 5'-ATAAA.
В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает добавление терминаторной полиТ-последовательности. В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает добавление терминаторной полиТ-последовательности в tracr- и/или парные tracr-последовательности. В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает добавление терминаторной полиТ-последовательности в направляющую последовательность. Терминаторная полиТ-последовательность может содержать 5 смежных T-оснований или более 5.
В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает изменение петель и/или "шпилек". В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает обеспечение минимум двух "шпилек" в направляющей последовательности. В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает обеспечение "шпильки", образованной при помощи комплементации между tracr- и парной tracr-последовательностью (прямой повтор). В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает обеспечение одной или нескольких дополнительных "шпилек" на 3'-конце последовательности tracrRNA или по направлению к нему. Например, "шпилька" может быть образована путем обеспечения самокомплементарных последовательностей в последовательности tracRNA, соединенных петлей так, что "шпилька" образуется при самосворачивании. В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает обеспечение дополнительных "шпилек", добавленных на 3' направляющей последовательности. В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает удлинение 5'-конца направляющей последовательности. В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает обеспечение одной или нескольких "шпилек" на 5'-конце направляющей последовательности. В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает введение последовательности (5'-AGGACGAAGTCCTAA) на 5'-конце направляющей последовательности. Другие последовательности, подходящие для образования "шпилек", известны специалисту в данной области, и их можно использовать в определенных аспектах настоящего изобретения. В некоторых аспектах настоящего изобретения предусмотрено по меньшей мере 2, 3, 4, 5 или более дополнительных "шпилек". В некоторых аспектах настоящего изобретения предусмотрено не более 10, 9, 8, 7, 6 дополнительных "шпилек". В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает две "шпильки". В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает три "шпильки". В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает самое большее пять "шпилек".
В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает обеспечение образования перекрестных связей или обеспечение одного или нескольких модифицированных нуклеотидов в полинуклеотидной последовательности. Модифицированные нуклеотиды и/или образование перекрестных связей могут предусматриваться в любой или во всех из tracr-, парных tracr- и/или направляющих последовательностей, и/или кодирующей фермент последовательности, и/или в векторных последовательностях. Модификации могут включать включение по меньшей мере одного не встречающегося в природе нуклеотида или модифицированного нуклеотида, или их аналогов. Модифицированные нуклеотиды могут быть модифицированы по фрагменту рибозы, фосфата и/или основания. Модифицированные нуклеотиды могут включать 2'-O-метил-аналоги, 2'-дезокси-аналоги или 2'-фтор-аналоги. Остов нуклеиновой кислоты можно модифицировать, например, можно использовать фосфотиоатный остов. Также возможно использование закрытых нуклеиновых кислот (LNA) или мостиковых нуклеиновых кислот (BNA). Дополнительные примеры модифицированных оснований включают без ограничения 2-аминопурин, 5-бромуридин, псевдоуридин, инозин, 7-метилгуанозин.
Будет понятно, что любая или все из вышеперечисленных модификаций могут быть предусмотрены в отдельности или в комбинации в данной системе CRISPR-Cas или ферментной системе CRISPR. Такая система может включать одну, две, три, четыре, пять или более указанных модификаций.
В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где фермент CRISPR является ферментом системы CRISPR II типа, к примеру, ферментом Cas9. В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где фермент CRISPR состоит менее чем из одной тысячи аминокислот, или менее чем из четырех тысяч аминокислот. В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где фермент Cas9 представляет собой StCas9 или StlCas9, или фермент Cas9 является ферментом Cas9 из организма, выбранного из группы, состоящей из рода Streptococcus, Campylobacter, Nitratifractor, Staphylococcus, Parvibaculum, Roseburia, Neisseria, Gluconacetobacter, Azospirillum, Sphaerochaeta, Lactobacillus, Eubacterium или Corynebacter. В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где фермент CRISPR является нуклеазой, управляющей расщеплением обеих нитей в определенной точке целевой последовательности.
В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где первый регуляторный элемент является промотором полимеразы III. В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где второй регуляторный элемент является промотором полимеразы II.
В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где направляющая последовательность содержит по меньшей мере пятнадцать нуклеотидов.
В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где модификация включает оптимизированную tracr-последовательность, и/или оптимизированную направляющую последовательность РНК, и/или совместно свернутую структуру tracr-последовательности и/или парной(ых) tracr-последовательности(ей), и/или стабилизирующие вторичные структуры tracr-последовательности, и/или tracr-последовательности с уменьшенным участком спаривания оснований, и/или tracr-последовательности, слитой с РНК-элементами; и/или в мультиплексной системе находятся две РНК, содержащие tracer и содержащие множество гидов, или одна РНК, содержащая множество химерных элементов.
В аспектах настоящего изобретения архитектура химерной РНК дополнительно оптимизирована в соответствии с результатами исследований мутационного процесса. В химерной РНК с двумя или более "шпильками" мутации в проксимальном прямом повторе для стабилизации "шпильки" могут привести к разрушению активности комплекса CRISPR. Мутации в дистальном прямом повторе для укорачивания или стабилизации "шпильки" могут не производить никакого воздействия на активность комплекса CRISPR. Рандомизация последовательности в петлевом участке между проксимальным и дистальным повторами может значительно снизить активность комплекса CRISPR. Замены одной пары оснований или рандомизация последовательности в линкерном участке между "шпильками" может привести к полной потере активности комплекса CRISPR. Стабилизация "шпильки" дистальных "шпилек", которые следуют за первой "шпилькой" после направляющей последовательности, может привести к сохранению или улучшению активности комплекса CRISPR. Соответственно, в предпочтительных вариантах осуществления настоящего изобретения архитектура химерной РНК может быть дополнительно оптимизирована путем получения меньшей химерной РНК, которая может иметь практическую значимость в отношении возможностей доставки для терапевтических целей и других применений, и этого можно достичь путем изменения дистального прямого повтора для того, чтобы укоротить или стабилизировать "шпильку". В дополнительных предпочтительных вариантах осуществления настоящего изобретения архитектура химерной РНК может быть дополнительно оптимизирована путем стабилизации одной или нескольких дистальных "шпилек". Стабилизация "шпилек" может включать модификацию последовательностей, подходящих для образования "шпилек". В некоторых аспектах настоящего изобретения предусмотрено по меньшей мере 2, 3, 4, 5 или более дополнительных "шпилек". В некоторых аспектах настоящего изобретения предусмотрено не более 10, 9, 8, 7, 6 дополнительных "шпилек". В некоторых аспектах настоящего изобретения стабилизацией может быть образование перекрестных связей и другие модификации. Модификации могут включать включение по меньшей мере одного не встречающегося в природе нуклеотида или модифицированного нуклеотида, или их аналогов. Модифицированные нуклеотиды могут быть модифицированы по фрагменту рибозы, фосфата и/или основания. Модифицированные нуклеотиды могут включать 2'-O-метил-аналоги, 2'-дезокси-аналоги или 2'-фтор-аналоги. Остов нуклеиновой кислоты можно модифицировать, например, можно использовать фосфотиоатный остов. Также возможно использование закрытых нуклеиновых кислот (LNA) или мостиковых нуклеиновых кислот (BNA). Дополнительные примеры модифицированных оснований включают без ограничения 2-аминопурин, 5-бромуридин, псевдоуридин, инозин, 7-метилгуанозин.
В одном аспекте настоящее изобретение предусматривает систему CRISPR-Cas или ферментную систему CRISPR, где фермент CRISPR кодон-оптимизирован для экспрессии в эукариотической клетке.
Соответственно, в некоторых аспектах настоящего изобретения длина tracRNA, необходимая для конструкции согласно настоящему изобретению, к примеру, химерной конструкции, необязательно должна быть фиксированной, и в некоторых аспектах настоящего изобретения она может составлять от 40 до 120 п.о., а в некоторых аспектах настоящего изобретения может составлять до полной длины tracr, к примеру, в некоторых аспектах настоящего изобретения, до 3'-конца tracr, которая прерывается сигналом терминации транскрипции в бактериальном геноме. В определенных вариантах осуществления длина tracRNA включает по меньшей мере нуклеотиды 1-67, а в некоторых вариантах осуществления по меньшей мере нуклеотиды 1-85 tracRNA дикого типа. В некоторых вариантах осуществления можно использовать по меньшей мере нуклеотиды, соответствующие нуклеотидам 1-67 или 1-85 tracRNA Cas9 S. pyogenes дикого типа. В тех случаях, когда в системе CRISPR используются ферменты, отличные от Cas9 или отличные от SpCas9, тогда в релевантной tracRNA дикого типа могут присутствовать соответствующие нуклеотиды. В некоторых вариантах осуществления длина tracRNA включает не более чем нуклеотиды 1-67 или 1-85 tracRNA дикого типа. В отношении оптимизации последовательности (к примеру, уменьшения полиТ-последовательностей), к примеру, касательно цепочек из T, внутренних по отношению к парной tracr (прямой повтор) или tracrRNA, в некоторых аспектах настоящего изобретения один или несколько T, присутствующих в полиТ-последовательности соответствующей последовательности дикого типа (то есть фрагмент из более 3, 4, 5, 6 или более смежных T-оснований; в некоторых вариантах осуществления фрагмент из не более 10, 9, 8, 7, 6 смежных T-оснований), могут быть заменены на отличный от T нуклеотид, к примеру, A, так что цепочка распадается на меньшие фрагменты из T, при этом каждый фрагмент имеет 4 или менее 4 (например, 3 или 2) смежных T. Если цепочка из T участвует в образовании "шпильки" (или структуры по типу "петля-на-стебле"), тогда предпочтительно, чтобы комплементарное основание для отличного от T основания было изменено на комплементарное отличному от T нуклеотиду. Например, если отличным от T основанием является A, тогда его комплементарное основание может быть изменено на T, к примеру, для сохранения или содействия сохранению вторичной структуры. К примеру, 5'-TTTTT может быть изменено с получением 5'-TTTAT, а комплементарная 5'-AAAAA может быть изменена на 5'-ATAAA. В отношении присутствия терминаторных полиТ-последовательностей в транскрипте tracr + парная tracr, к примеру, поли-T-терминатора (TTTTT или больше), в некоторых аспектах настоящего изобретения предпочтительно, чтобы таковой был добавлен к концу транскрипта, будь то в форме с двумя РНК (tracr и парной tracr) или с одной направляющей РНК. Касательно петель и "шпилек" в транскриптах tracr и парной tracr, в некоторых аспектах настоящего изобретения предпочтительно, чтобы минимум две "шпильки" присутствовали в химерной направляющей РНК. Первая "шпилька" может быть "шпилькой", образованной при помощи комплементации между tracr-последовательностью и парной tracr-последовательностью (прямой повтор). Вторая "шпилька" может быть на 3'-конце последовательности tracrRNA, и это может обеспечивать вторичную структуру для взаимодействия с Cas9. Дополнительные "шпильки" могут быть добавлены на 3' направляющей РНК, к примеру, в некоторых аспектах настоящего изобретения для того, чтобы повысить стабильность направляющей РНК. Кроме того, 5'-конец направляющей РНК в некоторых аспектах настоящего изобретения может быть удлинен. В некоторых аспектах настоящего изобретения можно рассматривать 20 п.о. на 5'-конце в качестве направляющей последовательности. 5'-часть может быть удлинена. Одна или несколько "шпилек" могут быть предусмотрены на 5'-части, к примеру, в некоторых аспектах настоящего изобретения это также может повышать стабильность направляющей РНК. В некоторых аспектах настоящего изобретения конкретная "шпилька" может быть обеспечена путем введения последовательности (5'-AGGACGAAGTCCTAA) на 5'-конце направляющей последовательности, а в некоторых аспектах настоящего изобретения это может помочь повысить стабильность. Другие последовательности, подходящие для образования "шпилек", известны специалисту в данной области, и их можно использовать в определенных аспектах настоящего изобретения. В некоторых аспектах настоящего изобретения предусмотрено по меньшей мере 2, 3, 4, 5 или более дополнительных "шпилек". В некоторых аспектах настоящего изобретения предусмотрено не более 10, 9, 8, 7, 6 дополнительных "шпилек". Вышеизложенное также предусматривает аспекты настоящего изобретения, включающие вторичную структуру в направляющие последовательности. В некоторых аспектах настоящего изобретения могут иметь место образование перекрестных связей и другие модификации, к примеру, для повышения стабильности. Модификации могут включать включение по меньшей мере одного не встречающегося в природе нуклеотида или модифицированного нуклеотида, или их аналогов. Модифицированные нуклеотиды могут быть модифицированы по фрагменту рибозы, фосфата и/или основания. Модифицированные нуклеотиды могут включать 2'-O-метил-аналоги, 2'-дезокси-аналоги или 2'-фтор-аналоги. Остов нуклеиновой кислоты можно модифицировать, например, можно использовать фосфотиоатный остов. Также возможно использование закрытых нуклеиновых кислот (LNA) или мостиковых нуклеиновых кислот (BNA). Дополнительные примеры модифицированных оснований включают без ограничения 2-аминопурин, 5-бромуридин, псевдоуридин, инозин, 7-метилгуанозин. Такие модификации или образование перекрестных связей могут иметь место в направляющей последовательности или других последовательностях, смежных с направляющей последовательностью.
Соответственно, целью настоящего изобретения не является охват в пределах настоящего изобретения любого ранее известного продукта, способа получения продукта или способа применения продукта, так что заявители оставляют за собой право и настоящим раскрывают отказ от прав на любой ранее известный продукт, процесс или способ. Следует дополнительно отметить, что настоящее изобретение не предназначено охватывать в пределах объема настоящего изобретения любой продукт, способ получения продукта или способ применения продукта, который не соответствует письменному описанию и требованиям достаточного раскрытия сути изобретения USPTO (первый пункт § 112 статьи 35 USC) или EPO (статья 83 EPC), так что заявители оставляют за собой право и настоящим раскрывают отказ от прав на любой ранее описанный продукт, способ получения продукта или способ применения продукта.
Следует отметить, что в данном раскрытии и особенно в формуле изобретения и/или параграфах такие выражения, как "содержит", "содержащийся", "содержащий" и т. п., могут иметь значение, приписываемое им в патентном законодательстве США, например, они могут означать "включает", "включенный", "включающий" и т. п., и что такие выражения, как "состоящий, по сути, из" и "состоит, по сути, из" имеют значение, приписываемое им в патентном законодательстве США, например, они допускают не указанные прямо элементы, но исключают элементы, которые имеются в известном уровне техники или которые влияют на основные или новые характеристики настоящего изобретения. Эти и другие варианты осуществления раскрыты или являются очевидными, исходя из следующего подробного описания, и охвачены им.
Краткое описание графических материалов
Новые признаки настоящего изобретения изложены с характерными особенностями в прилагаемой формуле изобретения. Лучшее понимание признаков и преимуществ настоящего изобретения будет доступно благодаря ссылке на следующее подробное описание, в котором изложены показательные варианты осуществления, в которых используют принципы настоящего изобретения, и на сопутствующие графические материалы.
На фигуре 1 изображена схематическая модель системы CRISPR. Нуклеаза Cas9 из Streptococcus pyogenes (желтый) нацелена на геномную ДНК при помощи синтетической направляющей РНК (sgRNA), состоящей из 20-нуклеотидной направляющей последовательности (голубой) и каркаса (красный). Направляющая последовательность образует пары оснований с ДНК-мишенью (голубой) непосредственно выше необходимого мотива, смежного с протоспейсером (PAM; пурпурный) 5'-NGG, и Cas9 опосредует двухцепочечный разрыв (DSB) на ~3 п.о. выше PAM (красный треугольник).
На фигурах 2A-F изображена показательная система CRISPR, возможный механизм действия, иллюстративная адаптация для экспрессии в эукариотических клетках и результаты тестов, оценивающих ядерную локализацию и активность CRISPR.
На фигурах 3A-C изображена показательная кассета экспрессии для экспрессии элементов системы CRISPR в эукариотических клетках, предсказанные структуры иллюстративных направляющих последовательностей и активность системы CRISPR, которая измерена в эукариотических и прокариотических клетках.
На фигурах 4A-D показаны результаты оценивания специфичности SpCas9 в отношении иллюстративной мишени.
На фигурах 5A-G изображена показательная векторная система и результаты ее применения при управлении гомологичной рекомбинацией в эукариотических клетках.
На фигурах 6A-C показано сравнение различных транскриптов tracrRNA для опосредованного Cas9 целенаправленного воздействия на ген.
На фигурах 7A-D изображена показательная система CRISPR, иллюстративная адаптация для экспрессии в эукариотических клетках и результаты тестов, оценивающих активность CRISPR.
На фигурах 8A-C изображены иллюстративные манипуляции с системой CRISPR для целенаправленного воздействия на локусы генома в клетках млекопитающего.
На фигурах 9A-B показаны результаты анализа с помощью нозерн-блоттинга процессинга crRNA в клетках млекопитающего.
На фигурах 10A-C показаны схематическое изображение химерных РНК и результаты анализа с помощью SURVEYOR относительно активности системы CRISPR в эукариотических клетках.
На фигурах 11A-B показано графическое изображение результатов анализа с помощью SURVEYOR относительно активности системы CRISPR в эукариотических клетках.
На фигуре 12 показаны предсказанные вторичные структуры для показательных химерных РНК, содержащих направляющую последовательность, парную tracr-последовательность и tracr-последовательность.
На фигуре 13 представлено филогенетическое дерево генов Cas.
На фигурах 14A-F показан филогенетический анализ, выявляющий пять семейств Cas9, включая три группы больших Cas9 (~1400 аминокислот) и две малых Cas9 (~1100 аминокислот).
На фигуре 15 показан график, показывающий функцию разных оптимизированных направляющих РНК.
На фигуре 16 показана последовательность и структура разных направляющих химерных РНК.
На фигуре 17 показана совместно свернутая структура tracrRNA и прямого повтора.
На фигуре 18 A и B показаны данные, полученные в результате in vitro оптимизации химерной направляющей РНК St1Cas9.
На фигуре 19A-B показано расщепление либо неметилированных, либо метилированных мишеней при помощи клеточного лизата с SpCas9.
На фигурах 20A-G показана оптимизация архитектуры направляющей РНК для SpCas9-опосредованного редактирования генома млекопитающих. (a) Схематическое изображение бицистронного вектора экспрессии (PX330) для управляемой промотором U6 одиночной направляющей РНК (sgRNA) и управляемого промотором CBh человеческого кодон-оптимизированного Cas9 Streptococcus pyogenes (hSpCas9), применяемых для всех последующих экспериментов. sgRNA, усеченная в разных указанных положениях, состоит из 20-нт направляющей последовательности (голубой) и каркаса (красный). (b) Анализ с помощью SURVEYOR в отношении опосредованных SpCas9 вставок/делеций в локусах EMX1 и PVALB человека. Стрелки указывают на ожидаемые фрагменты, полученные в результате расщепления с помощью SURVEYOR (n = 3). (c) Анализ с помощью нозерн-блоттинга для четырех усеченных архитектур sgRNA с U1 в качестве загрузочного контроля. (d) Как SpCas9 дикого типа (wt), так и никаза-мутант (D10A) SpCas9 способствовали вставке сайта HindIII в ген EMX1 человека. Однонитевые олигонуклеотиды (ssODN), ориентированные либо в смысловом, либо антисмысловом направлении по отношению к геномной последовательности, использовали в качестве матриц для гомологичной рекомбинации. (e) Схематическое изображение локуса SERPINB5 человека. sgRNA и PAM указаны при помощи цветных полос над последовательностями; метилцитозины (Me) выделены (розовый) и пронумерованы по отношению к сайту инициации транскрипции (TSS, +1). (f) Статус метилирования SERPINB5, оцененный при помощи бисульфитного секвенирования 16 клонов. Заполненные круги, метилированный CpG; белые круги, неметилированный CpG. (g) Эффективность модификации у трех sgRNA, нацеливающих на метилированный участок SERPINB5, оцененная при помощи "глубокого" секвинирования (n = 2). "Усы" указывают на интервалы Уилсона (способы он-лайн).
На фигурах 21A-B показана дополнительная оптимизация архитектуры sgRNA CRISPR-Cas. (a) Схематическое изображение четырех дополнительных архитектур sgRNA, I-IV. Каждая состоит из 20-нт направляющей последовательности (голубой), соединенной с прямым повтором (DR, серый), который гибридизируется с tracrRNA (красный). Гибрид DR-tracrRNA усечен в +12 или +22, которые указаны, искусственной структурой по типу "петля-на-стебле" GAAA. Положения усечения tracrRNA пронумерованы в соответствии с предварительно сообщенным сайтом инициации транскрипции для tracrRNA. Архитектуры II и IV sgRNA несут мутации в их полиU отрезках, которые могут служить в качестве терминаторов для осуществления преждевременной терминации транскрипции. (b) Анализ с помощью SURVEYOR в отношении опосредованных SpCas9 вставок/делеций в локусе EMX1 человека для целевых сайтов 1-3. Стрелки указывают на ожидаемые фрагменты, полученные в результате расщепления с помощью SURVEYOR (n = 3).
На фигуре 22 показана визуализация некоторых целевых сайтов в геноме человека.
На фигурах 23A-B показаны (A) схематическое изображение sgRNA и (B) анализ при помощи SURVEYOR пяти вариантов sgRNA касательно SaCas9 в отношении оптимальной усеченной архитектуры с наивысшей эффективностью расщепления.
Фигуры приведены в данном документе только в целях иллюстрации, и они не обязательно изображены в масштабе.
Подробное описание изобретения
Выражения “полинуклеотид”, “нуклеотид”, “нуклеотидная последовательность”, “нуклеиновая кислота” и “олигонуклеотид” используют взаимозаменяемо. Они обозначают полимерную форму нуклеотидов любой длины, как дезоксирибонуклеотидов, так и рибонуклеотидов или их аналогов. Полинуклеотиды могут обладать любой пространственной структурой и могут выполнять любую функцию, известную или неизвестную. Неограничивающими примерами полинуклеотидов являются следующие: кодирующие или некодирующе участки гена или фрагмента гена, локусы(локус), определенные в результате анализа сцепления, экзоны, интроны, информационная РНК (иРНК), транспортная РНК, рибосомная РНК, короткая интерферирующая РНК (siRNA), короткая шпилечная РНК (shRNA), микроРНК (miRNA), рибозимы, кДНК, рекомбинантные полинуклеотиды, разветвленные полинуклеотиды, плазмиды, векторы, выделенная ДНК любой последовательности, выделенная РНК любой последовательности, зонды для нуклеиновых кислот и праймеры. Полинуклеотид может содержать один или несколько модифицированных нуклеотидов, как, например, метилированные нуклеотиды и аналоги нуклеотидов. При наличии, модификации в нуклеотидную структуру могут быть внесены до или после сборки полимера. Последовательность нуклеотидов может прерываться отличными от нуклеотидов компонентами. Полинуклеотид можно дополнительно модифицировать после полимеризации, как, например, путем конъюгации с компонентом для мечения.
В аспектах настоящего изобретения выражения “химерная РНК”, “химерная направляющая РНК”, “направляющая РНК”, “одиночная направляющая РНК” и “синтетическая направляющая РНК” используют взаимозаменяемо, и они обозначают полинуклеотидную последовательность, содержащую направляющую последовательность, tracr-последовательность и парную tracr-последовательность. Выражение “направляющая последовательность” обозначает последовательность из приблизительно 20 п.о. в пределах направляющей РНК, которая определяет целевой сайт, и ее можно использовать взаимозаменяемо с выражениями “гид” или “спейсер”. Выражение “парная tracr-последовательность” также можно использовать взаимозаменяемо с выражением “прямой(ые) повтор(ы)”.
Используемое в данном документе выражение “дикий тип” является выражением из данной области, понятным специалисту в данной области, и означает типичную форму организма, штамма, гена или характеристики, которые встречаются в природе в отличие от мутантных или вариантных форм.
Используемое в данном документе выражение “вариант” следует понимать как означающее проявление качеств, которые характеризуются паттерном, который отличается от такового, встречающегося в природе.
Выражение “не встречающийся в природе” или “сконструированный” используют взаимозаменяемо, и оно указывает на вмешательство человека. Выражения, в тех случаях, когда они касаются молекул нуклеиновых кислот или полипептидов, означают, что молекула нуклеиновой кислоты или полипептид по меньшей мере практически не содержат по меньшей мере один отличный компонент, с которым они естественным образом связаны в природе и встречаются в природе.
“Комплементарность” означает способность нуклеиновой кислоты образовывать водородную(ые) связь(и) с другой последовательностью нуклеиновой кислоты при помощи либо традиционного спаривания оснований по Уотсону-Крику, либо других нетрадиционных типов. Процент комплементарности показывает процентную долю остатков в молекуле нуклеиновой кислоты, которые могут образовывать водородные связи (к примеру, образование пар по Уотсону-Крику) со второй последовательностью нуклеиновой кислоты (к примеру, при этом 5, 6, 7, 8, 9, 10 из 10 будут на 50%, 60%, 70%, 80%, 90% и 100% комплементарны). “Точная комплементарность” означает, что все граничащие остатки последовательности нуклеиновой кислоты будут связаны водородными связями с тем же количеством граничащих остатков во второй последовательности нуклеиновой кислоты. Выражение “практически комплементарный”, используемое в данном документе, означает степень комплементарности, которая составляет по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 97%, 98%, 99% или 100% в пределах участка из 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 30, 35, 40, 45, 50 или более нуклеотидов, или относится к двум нуклеиновым кислотам, которые гибиридизируются при жестких условиях.
Используемые в данном документе “жесткие условия” в отношении гибридизации означают условия, при которых нуклеиновая кислота с комплементарностью к целевой последовательности преимущественно гибридизируется с целевой последовательностью и практически не гибридизируется с нецелевыми последовательностями. Жесткие условия, как правило, являются зависимыми от последовательности и изменяются в зависимости от ряда факторов. Как правило, чем длиннее последовательность, тем выше температура, при которой последовательность специфично гибридизируется с целевой последовательностью. Неограничивающие примеры жестких условий описаны подробно в Tijssen (1993), Laboratory Techniques In Biochemistry And Molecular Biology-Hybridization With Nucleic Acid Probes Part I, Second Chapter “Overview of principles of hybridization and the strategy of nucleic acid probe assay”, Elsevier, N.Y.
“Гибридизация” означает реакцию, при которой один или несколько полинуклеотидов реагируют с образованием комплекса, который стабилизируется посредством образования водородных связей между основаниями нуклеотидных остатков. Образование водородных связей может происходить по принципу спаривания оснований по Уотсону-Крику, Хугстиновского связывания или любым другим специфичным к последовательности образом. Комплекс может содержать две нити, образующие дуплексную структуру, три или более нитей, образующих многонитевой комплекс, одиночную самогибридизирующуюся нить или любую их комбинацию. Реакция гибридизации может представлять собой стадию в более обширном способе, такую как начальная стадия ПЦР или расщепление полинуклеотида при помощи фермента. Последовательность, способную гибридизироваться с данной последовательностью, называют “комплементарной последовательностью” данной последовательности.
Используемое в данном документе выражение “стабилизация” или “повышение стабильности” в отношении компонентов системы CRISPR относится к предохранению или обеспечению устойчивости структуры молекулы. Это можно выполнить путем введения одной или нескольких мутаций, в том числе замен одной или нескольких пар оснований, увеличения количества "шпилек", образования перекрестных связей, разрушения определенных фрагментов нуклеотидов и других модификаций. Модификации могут включать включение по меньшей мере одного не встречающегося в природе нуклеотида или модифицированного нуклеотида, или их аналогов. Модифицированные нуклеотиды могут быть модифицированы по фрагменту рибозы, фосфата и/или основания. Модифицированные нуклеотиды могут включать 2'-O-метил-аналоги, 2'-дезокси-аналоги или 2'-фтор-аналоги. Остов нуклеиновой кислоты можно модифицировать, например, можно использовать фосфотиоатный остов. Также возможно использование закрытых нуклеиновых кислот (LNA) или мостиковых нуклеиновых кислот (BNA). Дополнительные примеры модифицированных оснований включают без ограничения 2-аминопурин, 5-бромуридин, псевдоуридин, инозин, 7-метилгуанозин. Такие модификации можно применять по отношению к любому компоненту системы CRISPR. В предпочтительных вариантах осуществления такие модификации осуществляют с РНК-компонентами, к примеру, направляющей РНК или химерной полинуклеотидной последовательностью.
Используемое в данном документе выражение “экспрессия” означает процесс, при котором полинуклеотид транскрибируется с ДНК-матрицы (как, например, с образованием мРНК или другого РНК-транскрипта), и/или способ, при помощи которого транскрибированная мРНК далее транслируется с образованием пептидов, полипептидов или белков. Транскрипты и кодируемые полипептиды можно в совокупности называть “продукт гена”. Если полинуклеотид получен из геномной ДНК, то экспрессия может включать сплайсинг иРНК в эукариотической клетке.
Выражения “полипептид”, “пептид” и “белок” используют взаимозаменяемо в данном документе для обозначения полимеров из аминокислот любой длины. Полимер может быть линейным или разветвленным, он может содержать модифицированные аминокислоты, и его структура может прерываться отличными от аминокислот компонентами. Выражения также охватывают полимер из аминокислот, который был модифицирован; например, образованием дисульфидных связей, гликозилированием, липидизацией, ацетилированием, фосфорилированием или любой другой манипуляцией, такой как соединение с компонентом для мечения. Используемое в данном документе выражение “аминокислота” включает природные и/или неприродные или синтетические аминокислоты, в том числе глицин и как D-, так и L-оптические изомеры, и аналоги аминокислот, и пептидомиметики.
Выражения “субъект”, “индивидуум” и “пациент” используют взаимозаменяемо в данном документе для обозначения позвоночного, предпочтительно млекопитающего, более предпочтительно человека. Млекопитающие включают без ограничения мышей, обезьян, людей, сельскохозяйственных животных, животных для спорта и домашних животных. Также охватываются ткани, клетки и их потомство биологического организма, полученные in vivo или культивированные in vitro. В некоторых вариантах осуществления субъектом может быть беспозвоночное животное, например, насекомое или нематода; в то время как в других субъектом может быть растение или гриб.
Выражения “терапевтическое средство”, “оказывающее терапевтический эффект средство” или “средство для лечения” используют взаимозаменяемо, и они означают молекулу или соединение, которые оказывают некоторое благоприятное воздействие при введении субъекту. Благоприятное воздействие включает осуществление диагностических определений; облегчение заболевания, симптома, нарушения или патологического состояния; ослабление или предупреждение начала проявления заболевания, симптома, нарушения или состояния; а также общее противодействие заболеванию, симптому, нарушению или патологическому состоянию.
Используемые в данном документе выражения “лечение”, или “осуществление лечения”, или “временное ослабление”, или “облегчение” используют взаимозаменяемо. Эти выражения означают подход для получения благоприятных или желательных результатов, в том числе без ограничения терапевтической полезности и/или профилактической полезности. Под терапевтическим эффектом понимают любые терапевтически значимые улучшение или действие в отношении одного или нескольких заболеваний, состояний или симптомов при лечении. Для профилактического эффекта композиции можно вводить субъекту с риском развития конкретного заболевания, состояния или симптома или субъекту, который сообщает об одном или нескольких физиологических симптомах заболевания, даже если заболевание, состояние или симптом могли еще не проявиться.
Выражение “эффективное количество” или “терапевтически эффективное количество” означает количество средства, которого достаточно для обеспечения благоприятных или желательных результатов. Терапевтически эффективное количество может изменяться в зависимости от одного или нескольких из: субъекта и болезненного состояния, которые подлежат лечению, веса и возраста субъекта, тяжести болезненного состояния, способа введения и подобного, что специалист в данной области легко может определить. Выражение также применимо к дозе, с помощью которой можно получить изображение для определения любым одним из способов визуализации, описанных в данном документе. Конкретная доза может изменяться в зависимости от одного или нескольких из: конкретного выбранного средства, режима дозирования, которому следуют, того, вводят ли его в комбинации с другими средствами, выбора времени введения, визуализируемой ткани и физической системы доставки, в которой оно заключено.
Практическое применение настоящего изобретения предусматривает, если не указано иное, традиционные методики иммунологии, биохимии, химии, молекулярной биологии, микробиологии, клеточной биологии, геномики и технологию рекомбинантной ДНК, которые находятся в пределах квалификации специалиста в данной области. См. Sambrook, Fritsch and Maniatis, MOLECULAR CLONING: A LABORATORY MANUAL, 2nd edition (1989); CURRENT PROTOCOLS IN MOLECULAR BIOLOGY (F. M. Ausubel, et al. eds., (1987)); серия METHODS IN ENZYMOLOGY (Academic Press, Inc.): PCR 2: A PRACTICAL APPROACH (M.J. MacPherson, B.D. Hames and G.R. Taylor eds. (1995)), Harlow and Lane, eds. (1988) ANTIBODIES, A LABORATORY MANUAL, and ANIMAL CELL CULTURE (R.I. Freshney, ed. (1987)).
Некоторые аспекты настоящего изобретения касаются векторных систем, содержащих один или несколько векторов, или векторов как таковых. Векторы могут быть разработаны для экспрессии транскриптов CRISPR (к примеру, транскриптов нуклеиновых кислот, белков или ферментов) в прокариотических или эукариотических клетках. Например, транскрипты CRISPR могут экспрессироваться в бактериальных клетках, как, например, Escherichia coli, клетках насекомых (с использованием бакуловирусных векторов экспрессии), клетках дрожжей или клетках млекопитающих. Подходящие клетки-хозяева дополнительно рассматриваются в Goeddel, GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990). В качестве альтернативы рекомбинантный вектор экспрессии может транскрибироваться и транслироваться in vitro, например, при помощи регуляторных последовательностей промотора T7 и полимеразы T7.
Векторы можно вводить и размножать в прокариоте. В некоторых вариантах осуществления прокариота используют для амплификации копий вектора, который предполагается вводить в эукариотическую клетку, или в качестве промежуточного вектора при получении вектора, который предполагается вводить в эукариотическую клетку (к примеру, путем амплификации плазмиды как части системы упаковки вирусного вектора). В некоторых вариантах осуществления прокариота используют для амплификации копий вектора и экспрессии одной или нескольких нуклеиновых кислот, как, например, для обеспечения источника одного или нескольких белков для доставки в клетку-хозяина или организм-хозяин. Экспрессию белков в прокариотах наиболее часто осуществляют в Escherichia coli с векторами, содержащими конститутивные или индуцибельные промоторы, управляющие экспрессией либо слитых белков, либо отличных от слитых белков. В слитых векторах добавляют некоторое количество аминокислот к белку, закодированному в них, как, например, к амино-концу рекомбинантного белка. Такие слитые векторы могут служить для одной или нескольких целей, как например: (i) для повышения экспрессии рекомбинантного белка; (ii) для повышения растворимости рекомбинантного белка и (iii) для содействия очистке рекомбинантного белка посредством функционирования в качестве лиганда при аффинной очистке. Часто в слитые векторы экспрессии сайт протеолитического расщепления вводят в место соединения фрагмента слияния и рекомбинантного белка для облегчения отделения рекомбинантного белка от фрагмента слияния после очистки слитого белка. Такие ферменты и их когнатные распознающие последовательности включают фактор Xa, тромбин и энтерокиназу. Иллюстративные слитые векторы экспрессии включают pGEX (Pharmacia Biotech Inc; Smith and Johnson, 1988. Gene 67: 31-40), pMAL (New England Biolabs, Беверли, Массачусетс) и pRIT5 (Pharmacia, Пискатауэй, Нью-Джерси), в которых глутатион-S-трансфераза (GST), мальтоза-связывающий белок E или белок A, соответственно, слиты с целевым рекомбинантным белком.
Примеры подходящих индуцибельных не являющихся слитыми векторов экспрессии E. coli включают pTrc (Amrann et al., (1988) Gene 69:301-315) и pET 11d (Studier et al., GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990) 60-89).
В некоторых вариантах осуществления вектор является вектором экспрессии для дрожжей. Примеры векторов для экспрессии в дрожжах Saccharomyces cerivisae включают pYepSec1 (Baldari, et al., 1987. EMBO J. 6: 229-234), pMFa (Kuijan and Herskowitz, 1982. Cell 30: 933-943), pJRY88 (Schultz et al., 1987. Gene 54: 113-123), pYES2 (Invitrogen Corporation, Сан-Диего, Калифорния) и picZ (InVitrogen Corp, Сан-Диего, Калифорния).
В некоторых вариантах осуществления вектор управляет экспрессией белка в клетках насекомых при помощи бакуловирусных векторов экспрессии. Бакуловирусные векторы, доступные для экспрессии белков в культивируемых клетках насекомых (к примеру, клетках SF9), включают группу pAc (Smith, et al., 1983. Mol. Cell. Biol. 3: 2156-2165) и группу pVL (Lucklow and Summers, 1989. Virology 170: 31-39).
В некоторых вариантах осуществления вектор способен управлять экспрессией одной или нескольких последовательностей в клетках млекопитающих при помощи вектора экспрессии для млекопитающих. Примеры векторов экспрессии для млекопитающих включают pCDM8 (Seed, 1987. Nature 329: 840) и pMT2PC (Kaufman, et al., 1987. EMBO J. 6: 187-195). При использовании клеток млекопитающих функции контроля вектора экспрессии, как правило, обеспечиваются одним или несколькими регуляторными элементами. Например, широко используемые промоторы получают из вируса полиомы, аденовируса 2, цитомегаловируса, вируса обезьян 40 и других, раскрытых в данном документе и известных в уровне техники. Что качается других подходящих систем экспрессии как для прокариотических, так и для эукариотических клеток, см., к примеру, главы 16 и 17 в Sambrook, et al., MOLECULAR CLONING: A LABORATORY MANUAL. 2nd ed., Cold Spring Harbor Laboratory, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y., 1989.
В некоторых вариантах осуществления рекомбинантные векторы экспрессии для млекопитающих способны управлять экспрессией нуклеиновой кислоты преимущественно в определенном типе клеток (к примеру, тканеспецифичные регуляторные элементы используют для экспрессии нуклеиновой кислоты). Тканеспецифичные регуляторные элементы известны из уровня техники. Неограничивающие примеры подходящих тканеспецифичных промоторов включают промотор гена альбумина (специфичный к печени; Pinkert, et al., 1987. Genes Dev. 1: 268-277), специфичные к лимфоидной ткани промоторы (Calame and Eaton, 1988. Adv. Immunol. 43: 235-275), в частности, промоторы рецепторов T-клеток (Winoto and Baltimore, 1989. EMBO J. 8: 729-733) и иммуноглобулины (Baneiji, et al., 1983. Cell 33: 729-740; Queen and Baltimore, 1983. Cell 33: 741-748), нейрон-специфичные промоторы (к примеру, промотор гена нейрофиламента; Byrne and Ruddle, 1989. Proc. Natl. Acad. Sci. USA 86: 5473-5477), специфичные к клеткам поджелудочной железы промоторы (Edlund, et al., 1985. Science 230: 912-916) и специфичные к клеткам молочной железы промоторы (к примеру, промотор молочной сыворотки; патент США № 4873316 и публикация европейской заявки № 264166). Регулируемые стадией развития промоторы также охвачены, к примеру, промоторы генов hox мыши (Kessel and Gruss, 1990. Science 249: 374-379) и промотор гена α-фетопротеина (Campes and Tilghman, 1989. Genes Dev. 3: 537-546).
В некоторых вариантах осуществления регуляторный элемент является функционально связанным с одним или несколькими элементами системы CRISPR так, чтобы управлять экспрессией одного или нескольких элементов системы CRISPR. В целом, CRISPR (короткие палиндромные повторы, регулярно расположенные группами), также известные как SPIDR (чередующиеся со спейсерами прямые повторы), составляют семейство локусов ДНК, которые, как правило, специфичны для определенного вида бактерий. Локус CRISPR включает определенный класс чередующихся коротких повторов последовательностей (SSR), которые были обнаружены у E. coli (Ishino et al., J. Bacteriol., 169:5429-5433 [1987]; и Nakata et al., J. Bacteriol., 171:3553-3556 [1989]), и ассоциированные гены. Подобные чередующиеся SSR были идентифицированы у Haloferax mediterranei, Streptococcus pyogenes, Anabaena и Mycobacterium tuberculosis (см., Groenen et al., Mol. Microbiol., 10:1057-1065 [1993]; Hoe et al., Emerg. Infect. Dis., 5:254-263 [1999]; Masepohl et al., Biochim. Biophys. Acta 1307:26-30 [1996]; и Mojica et al., Mol. Microbiol., 17:85-93 [1995]). Локусы CRISPR, как правило, отличаются от других SSR по структуре повторов, которые были названы короткими повторами с регулярными интервалами (SRSR) (Janssen et al., OMICS J. Integ. Biol., 6:23-33 [2002]; и Mojica et al., Mol. Microbiol., 36:244-246 [2000]). В целом, повторы являются короткими элементами, которые встречаются группами, которые регулярно разделены уникальными вставочными последовательностями с практически постоянной длинной (Mojica et al., [2000], выше). Несмотря на то, что последовательности повторов высоко консервативны между штаммами, некоторое количество чередующихся повторов и последовательностей спейсерных участков, как правило, отличаются от штамма к штамму (van Embden et al., J. Bacteriol., 182:2393-2401 [2000]). Локусы CRISPR были идентифицированы у более чем 40 видов прокариот (см., к примеру, Jansen et al., Mol. Microbiol., 43:1565-1575 [2002]; и Mojica et al., [2005]), в том числе, без ограничения, Aeropyrum, Pyrobaculum, Sulfolobus, Archaeoglobus, Halocarcula, Methanobacterium, Methanococcus, Methanosarcina, Methanopyrus, Pyrococcus, Picrophilus, Thermoplasma, Corynebacterium, Mycobacterium, Streptomyces, Aquifex, Porphyromonas, Chlorobium, Thermus, Bacillus, Listeria, Staphylococcus, Clostridium, Thermoanaerobacter, Mycoplasma, Fusobacterium, Azarcus, Chromobacterium, Neisseria, Nitrosomonas, Desulfovibrio, Geobacter, Myxococcus, Campylobacter, Wolinella, Acinetobacter, Erwinia, Escherichia, Legionella, Methylococcus, Pasteurella, Photobacterium, Salmonella, Xanthomonas, Yersinia, Treponema и Thermotoga.
В целом, “система CRISPR” означает в совокупности транскрипты и другие элементы, участвующие в экспрессии CRISPR-ассоциированных (“Cas”) генов или управлении их активностью, в том числе последовательности, кодирующие ген Cas, tracr-(транс-активируемую CRISPR) последовательность (к примеру, tracrRNA или активную частичную tracrRNA), парную tracr-последовательность (охватывающую “прямой повтор” и tracrRNA-процессированный неполный прямой повтор в контексте эндогенной системы CRISPR), направляющую последовательность (также называемую “спейсером” в контексте эндогенной системы CRISPR) или другие последовательности и транскрипты с локуса CRISPR. В некоторых вариантах осуществления один или несколько элементов системы CRISPR получены из системы CRISPR I типа, II типа или III типа. В некоторых вариантах осуществления один или несколько элементов системы CRISPR получены из определенного организма, содержащего эндогенную систему CRISPR, как, например, Streptococcus pyogenes. В целом, система CRISPR характеризуется элементами, которые способствуют образованию комплекса CRISPR на сайте целевой последовательности (также называемой протоспейсером в контексте эндогенной системы CRISPR). В контексте образования комплекса CRISPR “целевая последовательность” означает последовательность, по отношению к которой направляющая последовательность разработана так, чтобы обладать комплементарностью, где гибридизация между целевой последовательностью и направляющей последовательностью способствует образованию комплекса CRISPR. Полная комплементарность не обязательна при условии, что имеет место достаточная комплементарность для осуществления гибридизации и способствования образованию комплекса CRISPR. Целевая последовательность может содержать любой полинуклеотид, как, например, ДНК- или РНК-полинуклеотиды. В некоторых вариантах осуществления целевая последовательность расположена в ядре или цитоплазме клетки. В некоторых вариантах осуществления целевая последовательность может находиться в органелле эукариотической клетки, например, митохондрии или хлоропласте. Последовательность или матрицу, которую можно применять для рекомбинации в целевом локусе, содержащем целевые последовательности, называют “матрицей редактирования”, или “полинуклеотидом для редактирования”, или “последовательностью для редактирования”. В аспектах настоящего изобретения экзогенный матричный полинуклеотид можно называть матрицей редактирования. В одном аспекте настоящего изобретения рекомбинация является гомологичной рекомбинацией.
Как правило, в контексте эндогенной системы CRISPR образование комплекса CRISPR (содержащего направляющую последовательность, гибридизирующуюся с целевой последовательностью и образующую комплекс с одним или несколькими белками Cas) приводит к расщеплению одной или обеих нитей в или около (к примеру, в пределах 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 50 или более пар оснований от) целевой последовательности. Не желая быть связанными теорией, полагают, что tracr-последовательность, которая может содержать или состоять из всей или части tracr-последовательности дикого типа (к примеру, приблизительно или более чем приблизительно 20, 26, 32, 45, 48, 54, 63, 67, 85 или более нуклеотидов tracr-последовательности дикого типа), может также образовывать часть комплекса CRISPR, как, например, путем гибридизации вдоль по меньшей мере части tracr-последовательности со всей или частью парной tracr-последовательности, которая функционально связана с направляющей последовательностью. В некоторых вариантах осуществления tracr-последовательность обладает достаточной комплементарностью с парной tracr-последовательностью для гибридизации и участия в образовании комплекса CRISPR. Как и в случае с целевой последовательностью, полагают, что полная комплементарность не является необходимой при условии, что она является достаточной для выполнения функции. В некоторых вариантах осуществления tracr-последовательность характеризуется по меньшей мере 50%, 60%, 70%, 80%, 90%, 95% или 99% комплементарности последовательности по длине парной tracr-последовательности при оптимальном выравнивании. В некоторых вариантах осуществления один или несколько векторов, управляющих экспрессией одного или нескольких элементов системы CRISPR, вводят в клетку-хозяина, так что экспрессия элементов системы CRISPR управляет образованием комплекса CRISPR на одном или нескольких целевых сайтах. Например, каждое из фермента Cas, направляющей последовательности, связанной с парной tracr-последовательностью, и tracr-последовательности может быть функционально связано с отдельными регуляторными элементами в отдельных векторах. В качестве альтернативы, два или более элементов, экспрессируемых с одних и тех же или разных регуляторных элементов, можно объединять в одном векторе, с одним или несколькими дополнительными векторами, обеспечивая любые компоненты системы CRISPR, не включенные в первый вектор. Элементы системы CRISPR, которые объединены в один вектор, могут быть расположены в любой удобной ориентации, как, например, один элемент, расположенный 5' (“выше”) относительно или 3' (“ниже”) относительно второго элемента. Кодирующая последовательность одного элемента может быть расположена на той же или противоположной нити кодирующей последовательности второго элемента и направлена в том же или противоположном направлении. В некоторых вариантах осуществления один промотор управляет экспрессией транскрипта, кодирующего фермент CRISPR, и одной или нескольких из направляющей последовательности, парной tracr-последовательности (необязательно функционально связанной с направляющей последовательностью) и tracr-последовательности, встроенных в одну или несколько интронных последовательностей (к примеру, каждая в разном интроне, две или более по меньшей мере в одном интроне или все в одном интроне). В некоторых вариантах осуществления фермент CRISPR, направляющая последовательность, парная tracr-последовательность и tracr-последовательность функционально связаны с одним и тем же промотором и экспрессируются с него.
В некоторых вариантах осуществления вектор содержит один или несколько сайтов встраивания, как, например, последовательность узнавания рестрикционной эндонуклеазой (также называемая “сайтом клонирования”). В некоторых вариантах осуществления один или несколько сайтов встраивания (к примеру, приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более сайтов встраивания) расположены выше и/или ниже одного или нескольких элементов последовательности одного или нескольких векторов. В некоторых вариантах осуществления вектор содержит сайт встраивания выше парной tracr-последовательности и необязательно ниже регуляторного элемента, функционально связанного с парной tracr-последовательностью, так что после встраивания направляющей последовательности в сайт встраивания и при экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в эукариотической клетке. В некоторых вариантах осуществления вектор содержит два или более сайта встраивания, при этом каждый сайт встраивания расположен между двумя парными tracr-последовательностями с тем, чтобы обеспечить возможность встраивания направляющей последовательности в каждый сайт. В таком расположении две или более направляющие последовательности могут содержать две или более копий одной направляющей последовательности, две или более различных направляющих последовательностей или их комбинации. В тех случаях, когда применяют несколько различных направляющих последовательностей, может быть использована одна экспрессирующая конструкция для целенаправленного воздействия активности CRISPR на несколько различных соответствующих целевых последовательностей в клетке. Например, один вектор может содержать приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20 или более направляющих последовательностей. В некоторых вариантах осуществления приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более таких содержащих направляющие последовательности векторов могут быть предусмотрены и необязательно доставлены в клетку.
В некоторых вариантах осуществления вектор содержит регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей фермент CRISPR, как, например, белок Cas. Неограничивающие примеры белков Cas включают Cas1, Cas1B, Cas2, Cas3, Cas4, Cas5, Cas6, Cas7, Cas8, Cas9 (также известный как Csn1 и Csx12), Cas10, Csy1, Csy2, Csy3, Cse1, Cse2, Csc1, Csc2, Csa5, Csn2, Csm2, Csm3, Csm4, Csm5, Csm6, Cmr1, Cmr3, Cmr4, Cmr5, Cmr6, Csb1, Csb2, Csb3, Csx17, Csx14, Csx10, Csx16, CsaX, Csx3, Csx1, Csx15, Csf1, Csf2, Csf3, Csf4, их гомологи или их модифицированные варианты. Эти ферменты известны; например, аминокислотную последовательность белка Cas9 S. pyogenes можно найти в базе данных SwissProt под номером доступа Q99ZW2. В некоторых вариантах осуществления немодифицированный фермент CRISPR характеризуется активностью для расщепления ДНК, как, например, Cas9. В некоторых вариантах осуществления фермент CRISPR представляет собой Cas9, и им может быть Cas9 из S. pyogenes или S. pneumoniae. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или обеих нитей в определенной точке целевой последовательности, как, например, в пределах целевой последовательности и/или в пределах комплементарной последовательности целевой последовательности. В некоторых вариантах осуществления фермент CRISPR управляет расщеплением одной или обеих нитей в пределах приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 50, 100, 200, 500 или более пар оснований от первого или последнего нуклеотида целевой последовательности. В некоторых вариантах осуществления вектор кодирует фермент CRISPR, который является мутированным по отношению к соответствующему ферменту дикого типа, так что у мутированного фермента CRISPR отсутствует способность расщеплять одну или обе нити целевого полинуклеотида, содержащего целевую последовательность. Например, замена аспартата на аланин (D10A) в каталитическом домене RuvC I Cas9 из S. pyogenes трансформирует Cas9 из нуклеазы, которая расщепляет обе нити, в никазу (расщепляет одну нить). Другие примеры мутаций, которые превращают Cas9 в никазу, включают без ограничения H840A, N854A и N863A. В некоторых вариантах осуществления никазу Cas9 можно использовать в комбинации с направляющей(ими) последовательностью(ями), к примеру, двумя направляющими последовательностями, которые целенаправленно воздействуют, соответственно, на смысловую и антисмысловую нити ДНК-мишени. Эта комбинация позволяет надрезать обе нити и использовать их для индукции NHEJ. Авторы данной заявки показали (данные не показаны) эффективность двух мишеней для никаз (т. е. sgRNA, нацеленных на одну и ту же точку, но на различные нити ДНК) при индуцировании мутагенного NHEJ. Одиночная никаза (Cas9-D10A с одной sgRNA) не способна индуцировать NHEJ и создавать вставки/делеции, но авторы данной заявки показали, что двойная никаза (Cas9-D10A и две sgRNA, нацеленные на различные нити в одной и той же точке) способна делать это в эмбриональных стволовых клетках человека (hESC). Эффективность составляет приблизительно 50% таковой нуклеазы (т. е. нормального Cas9 без мутации D10) в hESC.
В качестве дополнительного примера два или более каталитических доменов Cas9 (RuvC I, RuvC II и RuvC III) можно подвергать мутациям с получением мутированного Cas9, у которого практически отсутствует вся активность для расщепления ДНК. В некоторых вариантах осуществления мутацию D10A объединяют с одной или несколькими из мутаций H840A, N854A или N863A с получением фермента Cas9, у которого практически отсутствует вся активность для расщепления ДНК. В некоторых вариантах осуществления фермент CRISPR рассматривают как такой, у которого практически отсутствует вся активность для расщепления ДНК, в случаях, когда активность для расщепления ДНК мутированного фермента составляет менее приблизительно 25%, 10%, 5%, 1%, 0,1%, 0,01% или меньше по отношению к его не мутированной форме. Могут быть целесообразными другие мутации; в тех случаях, когда Cas9 или другой фермент CRISPR получен из вида, отличного от S. pyogenes, могут быть произведены мутации в соответствующих аминокислотах для достижения подобных эффектов.
В некоторых вариантах осуществления кодирующая фермент последовательность, кодирующая фермент CRISPR, является кодон-оптимизированной для экспрессии в определенных клетках, как, например, эукариотических клетках. Эукариотические клетки могут быть клетками определенного организма или полученными из него, как, например, млекопитающего, в том числе, без ограничения, человека, мыши, крысы, кролика, собаки или отличного от человека примата. В целом, оптимизация кодонов означает способ модификации последовательности нуклеиновой кислоты для повышения экспрессии в представляющих интерес клетках-хозяевах путем замещения по меньшей мере одного кодона (к примеру, приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 10, 15, 20, 25, 50 или более кодонов) нативной последовательности кодонами, которые чаще или наиболее часто используют в генах этой клетки-хозяина, в то же время сохраняя нативную аминокислотную последовательность. Разные виды проявляют определенное "предпочтение" в отношении конкретных кодонов определенной аминокислоты. "Предпочтение" кодонов (различия в частоте использования кодонов между организмами) часто соотносят с эффективностью трансляции информационной РНК (иРНК), которая, в свою очередь, как полагают, зависит, среди прочего, от свойств кодонов, которые транслируются, и доступности конкретных молекул транспортной РНК (тРНК). Преобладание выбранных тРНК в клетке, как правило, является отражением кодонов, используемых наиболее часто при синтезе пептидов. Соответственно, гены могут быть приспособлены для оптимальной экспрессии генов в данном организме с использованием оптимизации кодонов. Таблицы частоты использования кодонов общедоступны, например, в "Базе данных частот использования кодонов" (“Codon Usage Database”), и эти таблицы можно адаптировать различными способами. См. Nakamura, Y., et al. “Codon usage tabulated from the international DNA sequence databases: status for the year 2000” Nucl. Acids Res. 28:292 (2000). Также доступны компьютерные алгоритмы для оптимизации кодонов определенной последовательности для экспрессии в определенной клетке-хозяине, как, например, также доступный Gene Forge (Aptagen; Джакобус, Пенсильвания). В некоторых вариантах осуществления один или несколько кодонов (к примеру, 1, 2, 3, 4, 5, 10, 15, 20, 25, 50 или более или все кодоны) в последовательности, кодирующей фермент CRISPR, соответствуют наиболее часто используемому кодону для определенной аминокислоты.
В некоторых вариантах осуществления вектор кодирует фермент CRISPR, содержащий одну или несколько последовательностей ядерной локализации (NLS), как, например, приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS. В некоторых вариантах осуществления фермент CRISPR содержит приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS на амино-конце или рядом с ним, приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS на карбокси-конце или рядом с ним или комбинацию этого (к примеру, одну или несколько NLS на амино-конце и одну или несколько NLS на карбокси-конце). В тех случаях, когда присутствуют несколько NLS, каждая может быть выбрана независимо от других, так что одна NLS может присутствовать в нескольких копиях и/или в комбинации с одной или несколькими другими NLS, присутствующими в одной или нескольких копиях. В предпочтительном варианте осуществления настоящего изобретения фермент CRISPR содержит самое большее 6 NLS. В некоторых вариантах осуществления считают, что NLS находится рядом с N- или C-концом в тех случаях, когда самая близкая аминокислота NLS находится в пределах приблизительно 1, 2, 3, 4, 5, 10, 15, 20, 25, 30, 40, 50 или более аминокислот вдоль полипетидной цепи от N- или C-конца. Обычно, NLS состоит из одной или нескольких коротких последовательностей положительно заряженных молекул лизина или аргинина, расположенных на поверхности белка, но известны другие типы NLS. Неограничивающие примеры NLS включают NLS-последовательности, полученные из: NLS из большого Т-антигена вируса SV40 с аминокислотной последовательностью PKKKRKV; NLS из нуклеоплазмина (к примеру, двойной NLS из нуклеоплазмина с последовательностью KRPAATKKAGQAKKKK); NLS из c-myc с аминокислотной последовательностью PAAKRVKLD или RQRRNELKRSP; NLS из hRNPA1 M9 с последовательностью NQSSNFGPMKGGNFGGRSSGPYGGGGQYFAKPRNQGGY; последовательность RMRIZFKNKGKDTAELRRRRVEVSVELRKAKKDEQILKRRNV домена IBB из импортина-альфа; последовательности VSRKRPRP и PPKKARED из Т-белка миомы; последовательность POPKKKPL из p53 человека; последовательность SALIKKKKKMAP из c-abl IV мыши; последовательности DRLRR и PKQKKRK из NS1 вируса гриппа; последовательность RKLKKKIKKL из дельта-антигена вируса гепатита; последовательность REKKKFLKRR из белка Mx1 мыши; последовательность KRKGDEVDGVDEVAKKKSKK из поли(АДФ-рибоза)-полимеразы человека и последовательность RKCLQAGMNLEARKTKK из рецепторов стероидных гормонов для глюкокортикоидов (человека).
В целом, одна или несколько NLS являются достаточно эффективными, чтобы управлять накоплением фермента CRISPR в обнаруживаемом количестве в ядре эукариотической клетки. В целом, степень проявления активности ядерной локализации может быть результатом следующего: количества NLS в ферменте CRISPR, конкретных(ой) используемых(ой) NLS или комбинации этих факторов. Обнаружение накопления в ядре можно выполнять при помощи любой подходящей методики. Например, детектируемый маркер может быть слит с ферментом CRISPR, так что расположение в клетке может быть визуализировано, как, например, в комбинации со средствами для обнаружения расположения ядра (к примеру, окрашивающим средством, специфичным к ядру, таким как DAPI). Примеры детектируемых маркеров включают флуоресцентные белки (такие как зеленые флуоресцентные белки или GFP; RFP; CFP) и эпитопные метки (HA-метку, flag-метку, SNAP-метку). Ядра клеток также можно выделять из клеток, содержимое которых можно затем анализировать при помощи любого подходящего способа для обнаружения белка, как, например, иммуногистохимии, вестерн-блоттинга или анализа на активность фермента. Накопление в ядре также можно определить опосредованно, как, например, при помощи анализа действия образования комплекса CRISPR (к примеру, анализа на расщепление ДНК или мутацию в целевой последовательности или анализа на измененную при помощи образования комплекса CRISPR и/или активности фермента CRISPR активность экспрессии генов) по сравнению с контролем, который не подвергали воздействию фермента или комплекса CRISPR или подвергали воздействию фермента CRISPR, у которого отсутствуют одна или несколько NLS.
В целом, направляющая последовательность представляет собой любую полинуклеотидную последовательность, обладающую достаточной комплементарностью с целевой полинуклеотидной последовательностью для гибридизации с целевой последовательностью и управления специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью. В некоторых вариантах осуществления степень комплементарности между направляющей последовательностью и ее соответствующей целевой последовательностью при оптимальном выравнивании с использованием подходящего алгоритма выравнивания составляет приблизительно или более чем приблизительно 50%, 60%, 75%, 80%, 85%, 90%, 95%, 97,5%, 99% или более. Оптимальное выравнивание можно определять при помощи любого подходящего алгоритма для выравниваемых последовательностей, неограничивающие примеры которого включают алгоритм Смита-Ватермана, алгоритм Нидлмана-Вунша, алгоритмы, основанные на преобразовании Барроуза-Уилера (к примеру, Burrows Wheeler Aligner), ClustalW, Clustal X, BLAT, Novoalign (Novocraft Technologies), ELAND (Illumina, Сан-Диего, Калифорния), SOAP (доступный на soap.genomics.org.cn) и Maq (доступный на maq.sourceforge.net). В некоторых вариантах осуществления направляющая последовательность составляет приблизительно или более чем приблизительно 5, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 75 или более нуклеотидов в длину. В некоторых вариантах осуществления направляющая последовательность составляет менее чем приблизительно 75, 50, 45, 40, 35, 30, 25, 20, 15, 12 или менее нуклеотидов в длину. Способность направляющей последовательности управлять специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью можно оценить при помощи любого подходящего анализа. Например, компоненты системы CRISPR, достаточные для образования комплекса CRISPR, в том числе направляющая последовательность, которую необходимо исследовать, могут быть доставлены в клетку-хозяина с соответствующей целевой последовательностью, как, например, при помощи трансфекции векторами, кодирующими компоненты последовательности CRISPR, с последующей оценкой предпочтительного расщепления в пределах целевой последовательности, как, например, при помощи анализа с помощью Surveyor, который описан в данном документе. Подобным образом, расщепление целевой полинуклеотидной последовательности может быть установлено в пробирке путем обеспечения целевой последовательности, компонентов комплекса CRISPR, в том числе направляющей последовательности, которую необходимо исследовать, и контрольной направляющей последовательности, отличной от тестовой направляющей последовательности, и сравнения воздействий тестовой и контрольной направляющей последовательности на связывание или скорость расщепления целевой последовательности. Другие анализы возможны и будут очевидны специалисту в данной области.
Направляющая последовательность может быть выбрана для целенаправленного воздействия на любую целевую последовательность. В некоторых вариантах осуществления целевая последовательность является последовательностью в пределах генома клетки. Иллюстративные целевые последовательности включают те, которые являются уникальными в целевом геноме. Например, для Cas9 S. pyogenes уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 в виде MMMMMMMMNNNNNNNNNNNNXGG, где NNNNNNNNNNNNXGG (N представляет собой A, G, T или C; и X может быть любым) характеризуется единичным появлением в геноме. Уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 S. pyogenes в виде MMMMMMMMMNNNNNNNNNNNXGG, где NNNNNNNNNNNXGG (N представляет собой A, G, T или C; и X может быть любым) характеризуется единичным появлением в геноме. Для Cas9 CRISPR1 S. thermophilus уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 в виде MMMMMMMMNNNNNNNNNNNNXXAGAAW, где NNNNNNNNNNNNXXAGAAW (N представляет собой A, G, T или C; X может быть любым; и W представляет собой A или T) характеризуется единичным появлением в геноме. Уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 CRISPR1 S. thermophilus в виде MMMMMMMMMNNNNNNNNNNNXXAGAAW, где NNNNNNNNNNNXXAGAAW (N представляет собой A, G, T или C; X может быть любым; и W представляет собой A или T) характеризуется единичным появлением в геноме. Для Cas9 S. pyogenes уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 в виде MMMMMMMMNNNNNNNNNNNNXGGXG, где NNNNNNNNNNNNXGGXG (N представляет собой A, G, T или C; и X может быть любым) характеризуется единичным появлением в геноме. Уникальная целевая последовательность в геноме может включать целевой сайт для Cas9 S. pyogenes в виде MMMMMMMMMNNNNNNNNNNNXGGXG, где NNNNNNNNNNNXGGXG (N представляет собой A, G, T или C; и X может быть любым) характеризуется единичным появлением в геноме. В каждой из этих последовательностей “M” может представлять собой A, G, T или C и не должен учитываться при идентификации последовательности как уникальной.
В некоторых вариантах осуществления направляющая последовательность выбрана для снижения доли вторичной структуры в направляющей последовательности. Вторичную структуру можно определить при помощи любого подходящего алгоритма сворачивания полинуклеотида. Некоторые программы основаны на вычислении минимальной свободной энергии Гиббса. Примером одного такого алгоритма является mFold, который описан Zuker и Stiegler (Nucleic Acids Res. 9 (1981), 133-148). Другим примером алгоритма сворачивания является доступный в режиме онлайн веб-сервер RNAfold, разработанный в Институте теоретической химии при Венском университете, в котором используется алгоритм прогнозирования структуры на основе центроидного метода (см., к примеру, A.R. Gruber et al.., 2008, Cell 106(1): 23-24; и PA Carr and GM Church, 2009, Nature Biotechnology 27(12): 1151-62). Дополнительные алгоритмы можно найти в заявке на патент США с серийным номером TBA (общая ссылка BI-2012/084 44790.11.2022); включенной в данный документ при помощи ссылки.
В общем, парная tracr-последовательность включает любую последовательность, которая характеризуется достаточной комплементарностью с tracr-последовательностью для содействия одному или нескольким из следующих: (1) вырезания направляющей последовательности, фланкированной парными tracr-последовательностями, в клетке, содержащей соответствующую tracr-последовательность; и (2) образования комплекса CRISPR на целевой последовательности, где комплекс CRISPR содержит парную tracr-последовательность, гибридизирующуюся с tracr-последовательностью. В общем, степень комплементарности указана на основании оптимального выравнивания парной tracr-последовательности и tracr-последовательности по длине более короткой из двух последовательностей. Оптимальное выравнивание можно определить при помощи любого подходящего алгоритма выравнивания и можно дополнительно высчитать для вторичных структур, как, например, самокомплементарность в пределах либо tracr-последовательности, либо парной tracr-последовательности. В некоторых вариантах осуществления степень комплементарности между tracr-последовательностью и парной tracr-последовательностью по длине более короткой из двух при оптимальном выравнивании составляет приблизительно или более чем приблизительно 25%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95%, 97,5%, 99% или более. Примерные иллюстрации оптимального выравнивания между tracr-последовательностью и парной tracr-последовательностью представлены на фигурах 12B и 13B. В некоторых вариантах осуществления tracr-последовательность составляет приблизительно или более чем приблизительно 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 25, 30, 40, 50 или более нуклеотидов в длину. В некоторых вариантах осуществления tracr-последовательность и парная tracr-последовательность содержатся в одном транскрипте, так что гибридизация между ними двумя дает транскрипт со вторичной структурой, такой как "шпилька". Предпочтительные петлеобразующие последовательности для использования в "шпилечных" структурах составляют четыре нуклеотида в длину и наиболее предпочтительно имеют последовательность GAAA. Однако можно использовать более короткие или длинные последовательности петли, а также альтернативные последовательности. Последовательности предпочтительно включают нуклеотидный триплет (например, AAA) и дополнительный нуклеотид (например, C или G). Примеры петлеобразующих последовательностей включают CAAA и AAAG. В одном варианте осуществления настоящего изобретения транскрипт или транскрибированная полинуклеотидная последовательность характеризуются по меньшей мере двумя или более "шпильками". В предпочтительных вариантах осуществления транскрипт характеризуется двумя, тремя, четырьмя или пятью "шпильками". В дополнительном варианте осуществления настоящего изобретения транскрипт характеризуется самое большее пятью "шпильками". В некоторых вариантах осуществления один транскрипт дополнительно включает последовательность терминации транскрипции; предпочтительно она является полиТ-последовательностью, например, из шести нуклеотидов T. Примерная иллюстрация такой "шпилечной" структуры представлена в нижней части фигуры 13B, где часть последовательности в 5' направлении по отношению к концевому “N” и выше петли соответствует парной tracr-последовательности, а часть последовательности в 3' направлении по отношению к петле соответствует tracr-последовательности. Дополнительными неограничивающими примерами отдельных полинуклеотидов, содержащих направляющую последовательность, парную tracr-последовательность и tracr-последовательность, являются следующие (перечисленные от 5' к 3'), где “N” представляет собой основание направляющей последовательности, первый блок букв нижнего регистра представляет собой парную tracr-последовательность, а второй блок букв нижнего регистра представляет собой tracr-последовательность, и конечная поли-T-последовательность представляет собой терминатор транскрипции: (1) NNNNNNNNNNNNNNNNNNNNgtttttgtactctcaagatttaGAAAtaaatcttgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgttttcgttatttaaTTTTTT; (2) NNNNNNNNNNNNNNNNNNNNgtttttgtactctcaGAAAtgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgttttcgttatttaaTTTTTT; (3) NNNNNNNNNNNNNNNNNNNNgtttttgtactctcaGAAAtgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgtTTTTTT; (4) NNNNNNNNNNNNNNNNNNNNgttttagagctaGAAAtagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgcTTTTTT; (5) NNNNNNNNNNNNNNNNNNNNgttttagagctaGAAATAGcaagttaaaataaggctagtccgttatcaacttgaaaaagtgTTTTTTT и (6) NNNNNNNNNNNNNNNNNNNNgttttagagctagAAATAGcaagttaaaataaggctagtccgttatcaTTTTTTTT. В некоторых вариантах осуществления последовательности (1)-(3) используют в комбинации с Cas9 из CRISPR1 S. thermophilus. В некоторых вариантах осуществления последовательности (4)-(6) используют в комбинации с Cas9 из S. pyogenes. В некоторых вариантах осуществления tracr-последовательность является транскриптом, отдельным от транскрипта, содержащего парную tracr-последовательность (как, например, показанная в верхней части фигуры 13B).
В некоторых вариантах осуществления также предусмотрена матрица для рекомбинации. Матрица для рекомбинации может быть компонентом другого вектора, который описан в данном документе, может содержаться в отдельном векторе или предусматриваться в виде отдельного полинуклеотида. В некоторых вариантах осуществления матрица для рекомбинации разработана так, чтобы служить в качестве матрицы при гомологичной рекомбинации, как, например, в пределах или рядом с целевой последовательностью, надрезанной или расщепленной ферментом CRISPR, в качестве части комплекса CRISPR. Матричный полинуклеотид может быть любой подходящей длины, как, например, приблизительно или более чем приблизительно 10, 15, 20, 25, 50, 75, 100, 150, 200, 500, 1000 или более нуклеотидов в длину. В некоторых вариантах осуществления матричный полинуклеотид комплементарен части полинуклеотида, содержащего целевую последовательность. При оптимальном выравнивании матричный полинуклеотид может перекрываться с одним или несколькими нуклеотидами целевых последовательностей (к примеру, с приблизительно или более чем приблизительно 1, 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100 или более нуклеотидами). В некоторых вариантах осуществления при оптимальном выравнивании матричной последовательности и полинуклеотида, содержащего целевую последовательность, наиболее близкий нуклеотид матричного полинуклеотида находится в пределах приблизительно 1, 5, 10, 15, 20, 25, 50, 75, 100, 200, 300, 400, 500, 1000, 5000, 10000 или более нуклеотидов от целевой последовательности.
В некоторых вариантах осуществления фермент CRISPR является частью слитого белка, содержащего один или несколько доменов гетерологичного белка (к примеру, приблизительно или более чем приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более доменов в дополнение к ферменту CRISPR). Слитый белок, содержащий фермент CRISPR, может содержать любую дополнительную последовательность белка и необязательно линкерную последовательность между любыми двумя доменами. Примеры белковых доменов, которые могут быть слиты с ферментом CRISPR, включают, без ограничения, эпитопные метки, последовательности генов-репортеров и белковые домены с одним или несколькими из следующих видов активности: метилазной активности, деметилазной активности, активности для активации транскрипции, активности для репрессии транскрипции, активности фактора освобождения при транскрипции, активности для модификации гистонов, активности для расщепления ДНК и активности для связывания нуклеиновой кислоты. Неограничивающие примеры эпитопных меток включают гистидиновые (His) метки, V5-метки, FLAG-метки, метки гемагглютинина вируса гриппа (HA), Myc-метки, VSV-G-метки и тиоредоксиновые (Trx) метки. Примеры генов-репортеров включают, без ограничения, глутатион-S-трансферазу (GST), пероксидазу хрена (HRP), хлорамфеникол-ацетилтрансферазу (CAT), бета-галактозидазу, бета-глюкуронидазу, люциферазу, зеленый флуоресцентный белок (GFP), HcRed, DsRed, голубой флуоресцентный белок (CFP), желтый флуоресцентный белок (YFP) и автофлуорисцирующие белки, в том числе синий флуоресцентный белок (BFP). Фермент CRISPR может быть слит с последовательностью гена, кодирующей белок или фрагмент белка, которые связываются с молекулой ДНК или связываются с другими клеточными молекулами, в том числе, без ограничения, связывающим мальтозу белком (MBP), S-меткой, продуктами слияния Lex A и ДНК-связывающего домена (DBD), продуктами слияния GAL4 и ДНК-связывающего домена и продуктами слияния белка BP16 вируса простого герпеса (HSV). Дополнительные домены, которые могут образовывать часть слитого белка, содержащего фермент CRISPR, описаны в US20110059502, включенном в данный документ при помощи ссылки. В некоторых вариантах осуществления меченный фермент CRISPR используют для идентификации расположения целевой последовательности.
В некоторых аспектах настоящее изобретение предусматривает способы, включающие доставку одного или нескольких полинуклеотидов, как, например, или одного или нескольких векторов, которые описаны в данном документе, одного или нескольких их транскриптов и/или одного или нескольких белков, транскрибируемых с них, в клетку-хозяина. В некоторых аспектах настоящее изобретение дополнительно предусматривает клетки, полученные при помощи таких способов, и организмы (такие как животные, растения или грибы), содержащие такие клетки или полученные из них. В некоторых вариантах осуществления фермент CRISPR в комбинации с (и необязательно образующий комплекс с) направляющей последовательностью доставляют в клетку. Традиционные способы переноса генов с использованием вирусов и без использования вирусов можно применять для введения нуклеиновых кислот в клетки млекопитающих или целевые ткани. Такие способы можно использовать для введения нуклеиновых кислот, кодирующих компоненты системы CRISPR, в клетки в культуре и в организме-хозяине. Системы доставки на основе отличных от вирусных векторов включают ДНК-плазмиды, РНК (к примеру, транскрипт вектора, описанного в данном документе), "оголенную" нуклеиновую кислоту и нуклеиновую кислоту, образующую комплекс со средством доставки, как, например, липосому. Системы доставки на основе вирусного вектора включают ДНК- и РНК-вирусы, которые имеют либо эписомальный, либо интегрированный геномы после доставки в клетку. В отношении обзора процедур генной терапии см. Anderson, Science 256:808-813 (1992); Nabel & Felgner, TIBTECH 11:211-217 (1993); Mitani & Caskey, TIBTECH 11:162-166 (1993); Dillon, TIBTECH 11:167-175 (1993); Miller, Nature 357:455-460 (1992); Van Brunt, Biotechnology 6(10):1149-1154 (1988); Vigne, Restorative Neurology and Neuroscience 8:35-36 (1995); Kremer & Perricaudet, British Medical Bulletin 51(1):31-44 (1995); Haddada et al., в Current Topics in Microbiology and Immunology, Doerfler and Böhm (eds) (1995); и Yu et al., Gene Therapy 1:13-26 (1994).
Способы отличной от вирусной доставки нуклеиновых кислот включают липофекцию, нуклеофекцию, микроинъекцию, баллистическую трансфекцию, виросомы, липосомы, иммунолипосомы, поликатион или конъюгаты липид:нуклеиновая кислота, "оголенную" ДНК, искусственные вирионы и повышенное при помощи средства поглощение ДНК. Липофекция описана, например, в патентах США №№ 5049386, 4946787 и 4897355), и реагенты для липофекции реализуются в промышленных масштабах (к примеру, Transfectam™ и Lipofectin™). Катионные и нейтральные липиды, которые подходят для эффективной липофекции полинуклеотидов с узнаванием рецептора, включают таковые из Felgner, WO 91/17424; WO 91/16024. Доставка может осуществляться в клетки (к примеру, in vitro или ex vivo введение) или целевые ткани (к примеру, in vivo введение).
Получение комплексов липид:нуклеиновая кислота, в том числе целенаправленно воздействующих липосом, как, например, иммунолипидных комплексов, хорошо известно специалистам в данной области (см., к примеру, Crystal, Science 270:404-410 (1995); Blaese et al., Cancer Gene Ther. 2:291-297 (1995); Behr et al., Bioconjugate Chem. 5:382-389 (1994); Remy et al., Bioconjugate Chem. 5:647-654 (1994); Gao et al., Gene Therapy 2:710-722 (1995); Ahmad et al., Cancer Res. 52:4817-4820 (1992); патенты США №№ 4186183, 4217344, 4235871, 4261975, 4485054, 4501728, 4774085, 4837028 и 4946787).
При применении систем на основе РНК- и ДНК-вирусов для доставки нуклеиновых кислот используют тщательно разработанные способы обеспечения целенаправленного воздействия вируса на конкретные клетки в организме и перемещения полезных последовательностей вируса в ядро. Вирусные векторы можно вводить непосредственно пациентам (in vivo) или их можно использовать для обработки клеток in vitro и модифицированные клетки можно необязательно вводить пациентам (ex vivo). Традиционные системы на основе вирусов могут включать ретровирусные, лентивирусные, аденовирусные векторы, векторы на основе аденоассоциированного вируса и вируса простого герпеса для переноса генов. Интеграция в геном хозяина возможна со способами переноса генов на основе ретровируса, лентивируса и аденоассоциированного вируса, что часто приводит к длительной экспрессии встроенного трансгена. Кроме того, высокие показатели эффективности трансдукции наблюдали у многих различных типов клеток и целевых тканей.
Тропизм ретровирусов может быть изменен путем включения чужеродных белков оболочки с расширением возможной целевой популяции целевых клеток. Лентивирусные векторы являются ретровирусными векторами, которые способны трансфицировать или инфицировать неделящиеся клетки и, как правило, дают высокие вирусные титры. Выбор системы переноса генов на основе ретровирусов, таким образом, будет зависеть от целевой ткани. Ретровирусные векторы состоят из действующих в цис-положении длинных концевых повторов с упаковывающей способностью до 6-10 п.о. чужеродной последовательности. Минимальных действующих в цис-положении LTR достаточно для репликации и упаковки векторов, которые затем используют для интеграции терапевтического гена в целевую клетку с получением постоянной экспрессии трансгена. Широко применяемые ретровирусные векторы включают такие, которые основаны на вирусе лейкоза мышей (MuLV), вирусе лейкоза гиббонов (GaLV), вирусе иммунодефицита обезьян (SIV), вирусе иммунодефицита человека (HIV) и их комбинациях (см., к примеру, Buchscher et al., J. Virol. 66:2731-2739 (1992); Johann et al., J. Virol. 66:1635-1640 (1992); Sommnerfelt et al., Virol. 176:58-59 (1990); Wilson et al., J. Virol. 63:2374-2378 (1989); Miller et al., J. Virol. 65:2220-2224 (1991); PCT/US94/05700).
В применениях, в которых транзиентная экспрессия является предпочтительной, можно применять системы на основе аденовирусов. Векторы на основе аденовирусов способны проявлять очень высокую эффективность трансдукции во многих типах клеток и не требуют деления клеток. С такими векторами были получены высокие титры и уровни экспрессии. Такой вектор можно получать в больших количествах в относительно простой системе. Векторы на основе аденоассоциированного вируса (“AAV”) также можно использовать для трансдукции клеток целевыми нуклеиновыми кислотами, к примеру, при получении in vitro нуклеиновых кислот и пептидов, и для процедур генной терапии in vivo и ex vivo (см., к примеру, West et al., Virology 160:38-47 (1987); патент США № 4797368; WO 93/24641; Kotin, Human Gene Therapy 5:793-801 (1994); Muzyczka, J. Clin. Invest. 94:1351 (1994). Создание рекомбинантных AAV-векторов описано в ряде публикаций, в том числе в патенте США № 5173414; Tratschin et al., Mol. Cell. Biol. 5:3251-3260 (1985); Tratschin, et al., Mol. Cell. Biol. 4:2072-2081 (1984); Hermonat & Muzyczka, PNAS 81:6466-6470 (1984); и Samulski et al., J. Virol. 63:03822-3828 (1989).
Упаковывающие клетки, как правило, используют для получения вирусных частиц, которые способны инфицировать клетку-хозяина. Такие клетки включают клетки 293, которые упаковывают аденовирус, и клетки ψ2 или клетки PA317, которые упаковывают ретровирус. Вирусные векторы, используемые в генной терапии, как правило, создают путем получения линии клеток, которые упаковывают вектор на основе нуклеиновой кислоты в вирусную частицу. Векторы обычно содержат минимальные вирусные последовательности, необходимые для упаковки и последующей интеграции в хозяина, при этом другие вирусные последовательности замещены кассетой экспрессии для экспрессии полинуклеотида(ов). Отсутствующие вирусные функции, как правило, обеспечивают во вспомогательном объекте при помощи линии упаковывающих клеток. Например, AAV-векторы, применяемые в генной терапии, как правило, имеют только ITR-последовательности из генома AAV, которые необходимы для упаковки и интеграции в геном хозяина. Вирусная ДНК упакована в линии клеток, которая содержит вспомогательную плазмиду, кодирующую другие гены AAV, а именно rep и cap, но без ITR-последовательностей. Линия клеток также может быть инфицирована аденовирусом в качестве вируса-помощника. Вирус-помощник способствует репликации AAV-вектора и экспрессии генов AAV из вспомогательной плазмиды. Вспомогательная плазмида не упакована в значительном количестве в связи с отсутствием ITR-последовательностей. Контаминация аденовирусом может быть снижена, к примеру, при помощи тепловой обработки, к которой аденовирус более чувствителен, чем AAV. Дополнительные способы доставки нуклеиновых кислот в клетки известны специалистам в данной области. См., например, US20030087817, включенный в данный документ при помощи ссылки.
В некоторых вариантах осуществления клетка-хозяин транзиентно или не транзиентно трасфицирована одним или несколькими векторами, описанными в данном документе. В некоторых вариантах осуществления клетка трансфицирована так, как это в естественных условиях происходит у субъекта. В некоторых вариантах осуществления клетка, которую трансфицируют, взята от субъекта. В некоторых вариантах осуществления клетка получена из клеток, взятых от субъекта, как, например, линия клеток. Широкий спектр линий клеток для культуры тканей известен из уровня техники. Примеры линий клеток включают, без ограничения, C8161, CCRF-CEM, MOLT, mIMCD-3, NHDF, HeLa-S3, Huh1, Huh4, Huh7, HUVEC, HASMC, HEKn, HEKa, MiaPaCell, Panc1, PC-3, TF1, CTLL-2, C1R, Rat6, CV1, RPTE, A10, T24, J82, A375, ARH-77, Calu1, SW480, SW620, SKOV3, SK-UT, CaCo2, P388D1, SEM-K2, WEHI-231, HB56, TIB55, Jurkat, J45.01, LRMB, Bcl-1, BC-3, IC21, DLD2, Raw264.7, NRK, NRK-52E, MRC5, MEF, Hep G2, HeLa B, HeLa T4, COS, COS-1, COS-6, COS-M6A, эпителиальные клетки почки обезьяны BS-C-1, эмбриональные фибробласты мыши BALB/ 3T3, 3T3 Swiss, 3T3-L1, фетальные фибробласты человека 132-d5; фибробласты мыши 10.1, 293-T, 3T3, 721, 9L, A2780, A2780ADR, A2780cis, A172, A20, A253, A431, A-549, ALC, B16, B35, клетки BCP-1, BEAS-2B, bEnd.3, BHK-21, BR 293, BxPC3, C3H-10T1/2, C6/36, Cal-27, CHO, CHO-7, CHO-IR, CHO-K1, CHO-K2, CHO-T, CHO Dhfr -/-, COR-L23, COR-L23/CPR, COR-L23/5010, COR-L23/R23, COS-7, COV-434, CML T1, CMT, CT26, D17, DH82, DU145, DuCaP, EL4, EM2, EM3, EMT6/AR1, EMT6/AR10.0, FM3, H1299, H69, HB54, HB55, HCA2, HEK-293, HeLa, Hepa1c1c7, HL-60, HMEC, HT-29, Jurkat, клетки JY, клетки K562, Ku812, KCL22, KG1, KYO1, LNCap, Ma-Mel 1-48, MC-38, MCF-7, MCF-10A, MDA-MB-231, MDA-MB-468, MDA-MB-435, MDCK II, MDCK II, MOR/0.2R, MONO-MAC 6, MTD-1A, MyEnd, NCI-H69/CPR, NCI-H69/LX10, NCI-H69/LX20, NCI-H69/LX4, NIH-3T3, NALM-1, NW-145, линии клеток OPCN / OPCT, Peer, PNT-1A / PNT 2, RenCa, RIN-5F, RMA/RMAS, клетки Saos-2, Sf-9, SkBr3, T2, T-47D, T84, линию клеток THP1, U373, U87, U937, VCaP, клетки Vero, WM39, WT-49, X63, YAC-1, YAR и их трансгенные варианты. Линии клеток доступны из ряда источников, известных специалистам в данной области (см., к примеру, Американскую коллекцию типовых культур (ATCC) (Манассас, Вирджиния)). В некоторых вариантах осуществления клетку, трансфицированную одним или несколькими векторами, описанными в данном документе, используют для получения новой линии клеток, содержащей одну или несколько полученных из вектора последовательностей. В некоторых вариантах осуществления клетку, транзиентно трансфицированную компонентами системы CRISPR, которая описана в данном документе (как, например, путем транзиентной трансфекции одним или несколькими векторами или трансфекции РНК), и модифицированную при помощи активности комплекса CRISPR, используют для получения новой линии клеток, содержащей клетки, которые содержат модификацию, но у которых отсутствует любая другая экзогенная последовательность. В некоторых вариантах осуществления клетки, транзиентно или не транзиентно трансфицированные одним или несколькими векторами, описанными в данном документе, или линии клеток, полученные из таких клеток, использовали при оценивании одного или нескольких тестовых соединений.
В некоторых вариантах осуществления один или несколько векторов, описанных в данном документе, используют для получения отличного от человека трансгенного животного или трансгенного растения. В некоторых вариантах осуществления трансгенным животным является млекопитающее, как, например, мышь, крыса или кролик. В определенных вариантах осуществления организмом или субъектом является растение. В определенных вариантах осуществления организмом, или субъектом, или растением является водоросль. Способы получения трансгенных растений и животных известны в уровне техники и, как правило, начинаются со способа трансфекции клетки, такого как описанный в данном документе. Также представлены трансгенные животные, как и трансгенные растения, в частности, сельскохозяйственные культуры и водоросли. Трансгенное животное или растение могут быть полезными в других путях применения, помимо обеспечения модели заболевания. Они могут включать производство пищи или кормопроизводство посредством биосинтеза, например, белков, углеводов, питательных веществ или витаминов на более высоких уровнях, чем будет наблюдаться в обычных условиях у дикого типа. В этом отношении предпочтительными являются трансгенные растения, в особенности зернобобовые и клубнеплоды, и животные, в особенности млекопитающие, такие как крупный рогатый скот (коровы, овцы, козы и свиньи), но также домашняя птица и съедобные насекомые.
Трансгенные водоросли или другие растения, такие как рапс, могут быть особенно применимыми в производстве растительных масел или таких видов биотоплива, как, например, спирты (особенно метанол и этанол). Они могут быть сконструированы для синтеза или сверхсинтеза масла или спиртов на высоких уровнях для применения в масложировой или биотопливной промышленности.
В одном аспекте настоящее изобретение предусматривает способы модификации целевого полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает обеспечение связывания комплекса CRISPR с целевым полинуклеотидом для осуществления расщепления указанного целевого полинуклеотида с модификацией, таким образом, целевого полинуклеотида, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизирующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизируется с tracr-последовательностью.
В одном аспекте настоящее изобретение предусматривает способ модификации экспрессии полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает обеспечение связывания комплекса CRISPR с полинуклеотидом так, что указанное связывание приводит к повышенной или пониженной экспрессии указанного полинуклеотида; где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, гибридизирующейся с целевой последовательностью в указанном целевом полинуклеотиде, где указанная направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизируется с tracr-последовательностью.
С учетом недавних достижений в области геномики сельскохозяйственных культур возможность применения системы CRISPR-Cas для осуществления эффективных и экономичных редактирования генов и манипуляции с ними обеспечит возможность быстрого отбора и сравнения одиночных и мультиплексных генетических манипуляций для трансформирования таких геномов в отношении повышенного производства и улучшенных признаков. В связи с этим делается ссылка на патенты США и публикации патентов США: патент США № 6603061 - опосредованный агробактериями способ трансформации растений (Agrobacterium-Mediated Plant Transformation Method); патент США № 7868149 - последовательности генома растений и их применение (Plant Genome Sequences and Uses Thereof) и US 2009/0100536 - трансгенные растения с улучшенными агротехническими признаками (Transgenic Plants with Enhanced Agronomic Traits), все содержания и раскрытия каждого из которых включены в данный документ при помощи ссылки в полном объеме. При осуществлении на практике настоящего изобретения содержание и раскрытие Morrell et al “Crop genomics:advances and applications” Nat Rev Genet. 2011 Dec 29;13(2):85-96 также включены в данный документ при помощи ссылки в полном объеме. В преимущественном варианте осуществления настоящего изобретения систему CRISPR/Cas9 используют для конструирования микроводорослей (пример 14). Соответственно, в данном документе ссылка на клетки животных также может быть применима, с учетом необходимых изменений, по отношению к клеткам растений, если явно не следует иное.
В одном аспекте настоящее изобретение предусматривает способы модификации целевого полинуклеотида в эукариотической клетке, что может происходить in vivo, ex vivo или in vitro. В некоторых вариантах осуществления способ включает забор клетки или популяции клеток от человека, или отличного от человека животного, или растения (в том числе микроскопических водорослей) и модификацию клетки или клеток. Культивирование можно осуществлять на любой стадии ex vivo. Клетку или клетки можно даже повторно вводить отличному от человека животному или в растение (в том числе микроскопические водоросли).
В одном аспекте настоящее изобретение предусматривает наборы, содержащие любой один или несколько из элементов, раскрытых в приведенных выше способах и композициях. В некоторых вариантах осуществления набор содержит векторную систему и инструкции по применению набора. В некоторых вариантах осуществления векторная система содержит (a) первый регуляторный элемент, функционально связанный с парной tracr-последовательностью и одним или несколькими сайтами встраивания для встраивания направляющей последовательности выше парной tracr-последовательности, где при экспрессии направляющая последовательность управляет специфичным к последовательности связыванием комплекса CRISPR с целевой последовательностью в эукариотической клетке, где комплекс CRISPR содержит фермент CRISPR, образующий комплекс с (1) направляющей последовательностью, которая гибридизируется с целевой последовательностью, и (2) парной tracr-последовательностью, которая гибридизируется с tracr-последовательностью; и/или (b) второй регуляторный элемент, функционально связанный с кодирующей фермент последовательностью, кодирующей указанный фермент CRISPR, содержащий последовательность ядерной локализации. Элементы могут быть предоставлены отдельно или в комбинациях и могут быть предоставлены в любом подходящем контейнере, как, например, пузырьке, флаконе или пробирке. В некоторых вариантах осуществления набор включает инструкции на одном или нескольких языках, например, на более чем одном языке.
В некоторых вариантах осуществления набор содержит один или несколько реагентов для применения в способе, в котором используется один или несколько элементов, описанных в данном документе. Реагенты могут быть предоставлены в любом подходящем контейнере. Например, набор может предусматривать один или несколько реакционных буферов или буферов для хранения. Реагенты могут быть предоставлены в форме, которая применима в конкретном анализе, или в форме, которая предусматривает добавление одного или нескольких других компонентов перед применением (к примеру, в форме концентрата или лиофилизированной форме). Буфер может быть любым буфером, в том числе без ограничения буфером с карбонатом натрия, буфером с бикарбонатом натрия, боратным буфером, Tris-буфером, буфером MOPS, буфером HEPES и их комбинациями. В некоторых вариантах осуществления буфер является щелочным. В некоторых вариантах осуществления буфер имеет значение pH от приблизительно 7 до приблизительно 10. В некоторых вариантах осуществления набор содержит один или несколько олигонуклеотидов, соответствующих направляющей последовательности, для встраивания в вектор для того, чтобы имела место функциональная связь направляющей последовательности и регуляторного элемента. В некоторых вариантах осуществления набор содержит матричный полинуклеотид для гомологичной рекомбинации.
В одном аспекте настоящее изобретение предусматривает способы применения одного или нескольких элементов системы CRISPR. Комплекс CRISPR по настоящему изобретению обеспечивает эффективное средство модификации целевого полинуклеотида. Комплекс CRISPR по настоящему изобретению характеризуется большим разнообразием полезных свойств, включая модификацию (например, делецию, вставку, транслокацию, инактивацию, активацию) целевого полинуклеотида во множестве типов клеток. Комплекс CRISPR по настоящему изобретению как таковой имеет широкий спектр применений, к примеру, в генной терапии, скрининге лекарственных средств, диагностике и прогнозировании заболеваний. Иллюстративный комплекс CRISPR содержит фермент CRISPR, образующий комплекс с направляющей последовательностью, которая гибридизируется с целевой последовательностью в целевом полинуклеотиде. Направляющая последовательность связана с парной tracr-последовательностью, которая, в свою очередь, гибридизируется с tracr-последовательностью.
Целевым полинуклеотидом комплекса CRISPR может быть любой полинуклеотид, эндогенный или экзогенный по отношению к эукариотической клетке. Например, целевой полинуклеотид может быть полинуклеотидом, находящимся в ядре эукариотической клетки. Целевой полинуклеотид может быть последовательностью, кодирующей продукт гена (к примеру, белок), или некодирующей последовательностью (к примеру, регуляторным полинуклеотидом или избыточной ДНК). Не желая быть связанными теорией, полагают, что целевая последовательность должна быть ассоциирована с PAM (мотивом, смежным с протоспейсером); то есть короткой последовательностью, узнаваемой комплексом CRISPR. Определенные требования в отношении последовательности и длины PAM различаются в зависимости от применяемого фермента CRISPR, но PAM, как правило, является последовательностью в 2-5 пар оснований, смежной с протоспейсером (то есть целевой последовательности). Примеры последовательностей PAM приведены в разделе "Примеры" ниже, и специалист в данной области сможет выявить дополнительные последовательности PAM для применения с данным ферментом CRISPR.
Целевой полинуклеотид комплекса CRISPR может включать некоторое количество ассоциированных с заболеваниями генов и полинуклеотидов, а также ассоциированных с биохимическими путями проведения сигнала генов и полинуклеотидов, которые перечислены в предварительных заявках на патент США 61/736527 и 61/748427 с общей ссылкой BI-2011/008/WSGR, номер в реестре 44063-701.101, и BI-2011/008/WSGR, номер в реестре 44063-701.102, соответственно, обе озаглавленные "СИСТЕМЫ, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИЯ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ" (SYSTEMS METHODS AND COMPOSITIONS FOR SEQUENCE MANIPULATION), поданные 12 декабря 2012 г. и 2 января 2013 г., соответственно, содержания всех из которых включены в данный документ при помощи ссылки в полном объеме.
Примеры целевых полинуклеотидов включают последовательность, ассоциированную с биохимическими путями передачи сигнала, к примеру, ген или полинуклеотид, ассоциированные с биохимическими путями передачи сигнала. Примеры целевых полинуклеотидов включают ассоциированный с заболеванием ген или полинуклеотид. “Ассоциированный с заболеванием” ген или полинуклеотид означает любой ген или полинуклеотид, который обеспечивает продукты транскрипции или трансляции на отклоняющемся от нормы уровне или в отклоняющейся от нормы форме в клетках, полученных из пораженных заболеванием тканей, по сравнению с тканями или клетками контроля без заболевания. Это может быть ген, который начинает экспрессироваться при ненормально высоком уровне; это может быть ген, который начинает экспрессироваться при ненормально низком уровне, где измененная экспрессия коррелирует с появлением и/или развитием заболевания. Ассоциированный с заболеванием ген также означает ген, несущий мутацию(и) или генетическое изменение, который непосредственно ответственен или находится в неравновесном сцеплении с геном(ами), который(е) ответственен(ны) за этиологию заболевания. Транскрибируемые или транслируемые продукты могут быть известными или неизвестными и могут быть на нормальном уровне или на отклоняющемся от нормального уровне.
Примеры ассоциированных с заболеваниями генов и полинуклеотидов доступны от Института генетической медицины Маккьюсика-Натанса (McKusick-Nathans Institute of Genetic Medicine) при Университете Джонса Хопкинса (Johns Hopkins University) (Балтимор, Мэриленд) и Национального центра биотехнологической информации (National Center for Biotechnology Information) при Национальной библиотеке медицины (National Library of Medicine) (Бетесда, Мэриленд), доступных во всемирной сети Интернет.
Примеры ассоциированных с заболеваниями генов и полинуклеотидов перечислены в таблицах A и B. Конкретная информация в отношении заболеваний доступна от Института генетической медицины Маккьюсика-Натанса (McKusick-Nathans Institute of Genetic Medicine) при Университете Джонса Хопкинса (Johns Hopkins University) (Балтимор, Мэриленд) и Национального центра биотехнологической информации (National Center for Biotechnology Information), Национальной библиотеки медицины (National Library of Medicine) (Бетесда, Мэриленд), доступных во всемирной сети Интернет. Примеры ассоциированных с биохимическими путями передачи сигнала генов и полинуклеотидов перечислены в таблице C.
Мутации в этих генах и путях могут приводить к продуцированию несоответствующих белков или белков в несоответствующих количествах, которые воздействуют на функцию. Дополнительные примеры генов, заболеваний и белков, таким образом, включены при помощи ссылки из предварительных заявок на патент США 61/736527 и 61/748427. Такие гены, белки и пути могут быть целевым полинуклеотидом комплекса CRISPR.
Таблица A
НАРУШЕНИЯ
Таблица B
Таблица C
гликонеогенез
кислоты
Варианты осуществления настоящего изобретения также относятся к способам и композициям, связанным с нокаутированием генов, амплифицированием генов и репарацией конкретных мутаций, ассоциированных с нестабильностью ДНК-повторов и неврологическими нарушениями (Robert D. Wells, Tetsuo Ashizawa, Genetic Instabilities and Neurological Diseases, Second Edition, Academic Press, Oct 13, 2011 - Medical). Как было обнаружено, определенные аспекты последовательностей тандемных повторов ответственны за более двадцати заболеваний человека (New insights into repeat instability: role of RNA•DNA hybrids. McIvor EI, Polak U, Napierala M. RNA Biol. 2010 Sep-Oct;7(5):551-8). Система CRISPR-Cas может быть приспособлена для корректировки таких дефектов геномной нестабильности.
Дополнительный аспект настоящего изобретения относится к использованию системы CRISPR-Cas для корректирования дефектов в генах EMP2A и EMP2B, которые, как было обнаружено, ассоциированы с болезнью Лафора. Болезнь Лафора представляет собой аутосомно-рецессивное состояние, которое характеризуется прогрессирующей миоклонус-эпилепсией, которая может начинаться как эпилептический приступ в подростковом возрасте. Некоторые случаи заболевания могут вызываться мутациями в генах, которые уже были выявлены. Заболевание вызывает приступы, мышечные спазмы, затрудненную ходьбу, слабоумие и, в конечном итоге, смерть. В настоящее время не существует терапии, которая показала эффективность против развития заболевания. На другие генетические расстройства, ассоциированные с эпилепсией, также можно целенаправленно воздействовать при помощи системы CRISPR-Cas, и лежащая в основе генетика дополнительно описана в Genetics of Epilepsy and Genetic Epilepsies, edited by Giuliano Avanzini, Jeffrey L. Noebels, Mariani Foundation Paediatric Neurology:20; 2009).
В еще одном аспекте настоящего изобретения систему CRISPR-Cas можно использовать для корректировки офтальмологических дефектов, которые являются результатом нескольких генетических мутаций, дополнительно описанных в Genetic Diseases of the Eye, Second Edition, edited by Elias I. Traboulsi, Oxford University Press, 2012.
Некоторые дополнительные аспекты настоящего изобретения связаны с корректированием дефектов, ассоциированных с широким спектром генетических заболеваний, которые дополнительно описаны на веб-сайте Национальных институтов здравоохранения (National Institutes of Health) в тематическом подразделе "Наследственные заболевания" ("Genetic Disorders"). Наследственные заболевания головного мозга могут включать без ограничения адренолейкодистрофию, агенезию мозолистого тела, синдром Айкарди, синдром Альперса, болезнь Альцгеймера, синдром Барта, болезнь Баттена, CADASIL, мозжечковую дегенерацию, болезнь Фабри, синдром Герстмана-Штраусслера-Шейнкера, болезнь Гентингтона и другие связанные с триплетным повтором нарушения, болезнь Лея, синдром Леша-Найхана, болезнь Менкеса, типы митохондриальной миопатии и кольпоцефалию по критериям NINDS. Такие заболевания дополнительно описаны на веб-сайте Национальных институтов здравоохранения (National Institutes of Health) в тематическом подразделе "Наследственные заболевания головного мозга" ("Genetic Brain Disorders").
В некоторых вариантах осуществления состоянием может быть неоплазия. В некоторых вариантах осуществления, где состоянием является неоплазия, гены, на которые целенаправленно воздействуют, являются любыми из перечисленных в таблице A (в данном случае PTEN и так далее). В некоторых вариантах осуществления состоянием может быть возрастная дегенерация желтого пятна. В некоторых вариантах осуществления состоянием может быть шизофреническое нарушение. В некоторых вариантах осуществления состоянием может быть связанное с тринуклеотидным повтором нарушение. В некоторых вариантах осуществления состоянием может быть синдром ломкой X-хромосомы. В некоторых вариантах осуществления состоянием может быть связанное с активностью секретазы нарушение. В некоторых вариантах осуществления состоянием может быть связанное с прионами нарушение. В некоторых вариантах осуществления состоянием может быть ALS. В некоторых вариантах осуществления состоянием может быть привыкание к наркотическим средствам. В некоторых вариантах осуществления состоянием может быть аутизм. В некоторых вариантах осуществления состоянием может быть болезнь Альцгеймера. В некоторых вариантах осуществления состоянием может быть воспаление. В некоторых вариантах осуществления состоянием может быть болезнь Паркинсона.
Примеры белков, ассоциированных с болезнью Паркинсона, включают без ограничения α-синуклеин, DJ-1, LRRK2, PINK1, паркин, UCHL1, синфилин-1 и NURR1.
Примеры связанных с привыканием белков могут включать, например, ABAT.
Примеры связанных с воспалением белков могут включать, например, моноцитарный хемоаттрактантный белок-1 (monocyte chemoattractant protein-1) (MCP1), кодируемый геном Ccr2, C-C рецептор хемокина 5 типа (C-C chemokine receptor type 5) (CCR5), кодируемый геном Ccr5, IgG-рецептор IIB (IgG receptor IIB) (FCGR2b, также называемый CD32), кодируемый геном Fcgr2b, или белок Fc-эпсилон-R1g (Fc epsilon R1g) (FCER1g), кодируемый геном Fcer1g.
Примеры ассоциированных с заболеваниями сердечно-сосудистой системы белков могут включать, например, IL1B (интерлейкин 1, бета (interleukin 1, beta)), XDH (ксантиндегидрогеназу (xanthine dehydrogenase)), TP53 (опухолевый белок p53 (tumor protein p53)), PTGIS (простагландин-I2(простациклин)-синтазу (prostaglandin I2 (prostacyclin) synthase)), MB (миоглобин (myoglobin)), IL4 (интерлейкин 4 (interleukin 4)), ANGPT1 (ангиопоэтин 1 (angiopoietin 1)), ABCG8 (АТФ-связывающую кассету, подсемейство G (WHITE), представитель 8 (ATP-binding cassette, sub-family G (WHITE), member 8)) или CTSK (катепсин K (cathepsin K)).
Примеры ассоциированных с болезнью Альцгеймера белков могут включать, например, белок, представляющий собой рецептор липопротеинов очень низкой плотности (very low density lipoprotein receptor protein) (VLDLR), кодируемый геном VLDLR, убиквитин-подобный модификатор-активирующий фермент 1 (ubiquitin-like modifier activating enzyme 1) (UBA1), кодируемый геном UBA1, или белок, являющийся каталитической субъединицей NEDD8-активирующего фермента E1 (NEDD8-activating enzyme E1 catalytic subunit protein) (UBE1C), кодируемый геном UBA3.
Примеры белков, ассоциированных с расстройствами аутистического спектра, могут включать, например, белок 1, ассоциированный с периферическим бензодиазепиновым рецептором (benzodiazapine receptor (peripheral) associated protein 1) (BZRAP1), кодируемый геном BZRAP1, белок, представитель 2 семейства AF4/FMR2 (AF4/FMR2 family member 2 protein) (AFF2), кодируемый геном AFF2 (также называемый MFR2), белок-аутосомный гомолог 1, ассоциированный с умственной отсталостью, связанной с ломкой X-хромосомой (fragile X mental retardation autosomal homolog 1 protein) (FXR1), кодируемый геном FXR1, или белок-аутосомный гомлог 2, ассоциированный с умственной отсталостью, связанной с ломкой X-хромосомой (fragile X mental retardation autosomal homolog 2 protein) (FXR2), кодируемый геном FXR2.
Примеры белков, ассоциированных с дегенерацией желтого пятна, могут включать, например, АТФ-связывающую кассету, белок-представитель 4 подсемейства A (ABC1) (ATP-binding cassette, sub-family A (ABC1) member 4 protein) (ABCA4), кодируемый геном ABCR, белок-аполипротеин E (apolipoprotein E protein) (APOE), кодируемый геном APOE, или белок-лиганд 2 хемокина (C-C мотив) (chemokine (C-C motif) Ligand 2 protein) (CCL2), кодируемый геном CCL2.
Примеры белков, ассоциированных с шизофренией, могут включать NRG1, ErbB4, CPLX1, TPH1, TPH2, NRXN1, GSK3A, BDNF, DISC1, GSK3B и их комбинации.
Примеры белков, вовлеченных в подавление опухоли, могут включать, например, ATM (мутированный, атаксия-телеангиэктазия (ataxia telangiectasia mutated)), ATR (атаксия-телеангиэктазия- и Rad3-родственный (ataxia telangiectasia and Rad3 related)), EGFR (рецептор эпидермального фактора роста (epidermal growth factor receptor)), ERBB2 (гомолог 2 v-erb-b2 эритробластического лейкоза вирусного онкогена (v-erb-b2 erythroblastic leukemia viral oncogene homolog 2)), ERBB3 (гомолог 3 v-erb-b2 эритробластического лейкоза вирусного онкогена (v-erb-b2 erythroblastic leukemia viral oncogene homolog 3)), ERBB4 (гомолог 4 v-erb-b2 эритробластического лейкоза вирусного онкогена (v-erb-b2 erythroblastic leukemia viral oncogene homolog 4)), Notch 1, Notch2, Notch 3 или Notch 4.
Примеры белков, ассоциированных с нарушением, связанным с активностью секретазы, могут включать, например, PSENEN (presenilin enhancer 2 homolog (C. elegans)), CTSB (катепсин B (cathepsin B)), PSEN1 (пресенилин 1 (presenilin 1)), APP (белок-предшественник бета-амилоида (A4) (amyloid beta (A4) precursor protein)), APH1B (anterior pharynx defective 1 homolog B (C. elegans)), PSEN2 (пресенилин 2 (болезнь Альцгеймера 4) (presenilin 2 (Alzheimer disease 4)) или BACE1 (APP-расщепляющий фермент 1 по бета-сайту (beta-site APP-cleaving enzyme 1)).
Примеры белков, ассоциированных с амиотрофическим латеральным склерозом, могут включать, например, SOD1 (супероксиддисмутазу 1 (superoxide dismutase 1)), ALS2 (белок, ассоциированный с амиотрофическим латеральным склерозом 2 (amyotrophic lateral sclerosis 2)), FUS (РНК-связывающий белок FUS (fused in sarcoma)), TARDBP (TAR-ДНК связывающий белок (TAR DNA binding protein)), VAGFA (фактор роста эндотелия сосудов A (vascular endothelial growth factor A)), VAGFB (фактор роста эндотелия сосудов B (vascular endothelial growth factor B)) и VAGFC (фактор роста эндотелия сосудов C (vascular endothelial growth factor C)) и любую их комбинацию.
Примеры белков, ассоциированных с прионными болезнями, могут включать SOD1 (супероксиддисмутазу 1), ALS2 (белок, ассоциированный с амиотрофическим латеральным склерозом 2 (amyotrophic lateral sclerosis 2)), FUS (РНК-связывающий белок FUS (fused in sarcoma)), TARDBP (TAR-ДНК связывающий белок (TAR DNA binding protein)), VAGFA (фактор роста эндотелия сосудов A (vascular endothelial growth factor A)), VAGFB (фактор роста эндотелия сосудов B (vascular endothelial growth factor B)) и VAGFC (фактор роста эндотелия сосудов C (vascular endothelial growth factor C)) и любую их комбинацию.
Примеры белков, связанных с нейродегенеративными состояниями при прионных болезнях, могут включать, например, A2M (альфа-2-макроглобулин (Alpha-2-Macroglobulin)), AATF (фактор транскрипции, противодействующий апоптозу (Apoptosis antagonizing transcription factor)), ACPP (простатоспецифическую кислую фосфатазу (Acid phosphatase prostate)), ACTA2 (альфа-актин 2 гладкой мускулатуры аорты (Actin alpha 2 smooth muscle aorta)), ADAM22 (ADAM, металлопептидазный домен (ADAM metallopeptidase domain)), ADORA3 (аденозиновый рецептор A3 типа (Adenosine A3 receptor)) или ADRA1D (альфа-1D адренергический рецептор для альфа-1D адренорецептора (Alpha-1D adrenergic receptor for Alpha-1D adrenoreceptor)).
Примеры белков, ассоциированных с иммунодефицитом, могут включать, например, A2M [альфа-2-макроглобулин (alpha-2-macroglobulin)]; AANAT [арилалкиламин-N-ацетилтрансферазу (arylalkylamine N-acetyltransferase)]; ABCA1 [АТФ-связывающую кассету, подсемейство A (ABC1), представитель 1 (ATP-binding cassette, sub-family A (ABC1), member 1)]; ABCA2 [АТФ-связывающую кассету, подсемейство A (ABC1), представитель 2 (ATP-binding cassette, sub-family A (ABC1), member 2)] или ABCA3 [АТФ-связывающую кассету, подсемейство A (ABC1), представитель 3 (ATP-binding cassette, sub-family A (ABC1), member 3)].
Примеры белков, ассоциированных с нарушениями, связанными с тринуклеотидным повтором, включают, например, AR (андрогеновый рецептор (androgen receptor)), FMR1 (белок 1, ассоциированный с умственной отсталостью, связанной с ломкой X-хромосомой (fragile X mental retardation 1)), HTT (хантигтин (huntingtin)) или DMPK (протеинкиназу, ассоциированную с мышечной дистрофией (dystrophia myotonica-protein kinase)), FXN (фратаксин (frataxin)), ATXN2 (атаксин 2 (ataxin 2)).
Примеры белков, ассоциированных с нарушениями передачи нервных импульсов включают, например, SST (соматостатин (somatostatin)), NOS1 (синтазу оксида азота 1 (нейрональную) (nitric oxide synthase 1 (neuronal)), ADRA2A (адренергический, альфа-2A-, рецептор (adrenergic, alpha-2A-, receptor)), ADRA2C (адренергический, альфа-2C-, рецептор (adrenergic, alpha-2C-, receptor)), TACR1 (тахикининовый рецептор 1 (tachykinin receptor 1)) или HTR2c (5-гидрокситриптаминовый (серотониновый) рецептор 2C (5-hydroxytryptamine (serotonin) receptor 2C)).
Примеры последовательностей, ассоциированных с неврологическим развитием, включают, например, A2BP1 [атаксин 2-связывающий белок 1 (ataxin 2-binding protein 1)], AADAT [аминоадипатаминотрансферазу (aminoadipate aminotransferase)], AANAT [арилалкиламин-N-ацетилтрансферазу (arylalkylamine N-acetyltransferase)], ABAT [4-аминобутиратаминтрансферазу (4-aminobutyrate aminotransferase)], ABCA1 [АТФ-связывающую кассету, подсемейство A (ABC1), представитель 1 (ATP-binding cassette, sub-family A (ABC1), member 1)] или ABCA13 [АТФ-связывающую кассету, подсемейство A (ABC1), представитель 13 (ATP-binding cassette, sub-family A (ABC1), member 13)].
Дополнительные примеры предпочтительных состояний, которые подлежат лечению с помощью данной системы, включают те, которые могут быть выбраны из синдрома Айкарди-Гутьереса; болезни Александера; синдрома Аллана-Херндона-Дадли; связанных с геном POLG нарушений; альфа-маннозидоза (II и III тип); синдрома Альстрема; синдрома Ангельмана; атаксии-телеангиэктазии; нейронного высоковидного липофусциноза; бета-талассемии; двусторонней атрофии зрительного нерва и (инфантильной) атрофии зрительного нерва 1 типа; ретинобластомы (двусторонней); болезни Канавана; церебро-окуло-фацио-скелетного синдрома 1 [COFS1]; церебротендинального ксантоматоза; синдрома Корнелии де Ланге; связанных с геном MAPT нарушений; наследственных прионных болезней; синдрома Драве; семейной болезни Альцгеймера с ранним началом; атаксии Фридрейха [FRDA]; синдрома Фринса; фукозидоза; врожденной мышечной дистрофии Фукуямы; галактосиалидоза; болезни Гоше; органической ацидемии; гемофагоцитарного лимфогистиоцитоза; синдрома прогерии Гетчинсона-Гилфорда; муколипидоза II; инфантильной болезни накопления свободной сиаловой кислоты; ассоциированной с геном PLA2G6 нейродегенерации; синдрома Джервелла-Ланге-Нильсена; узелкового врожденного буллезного эпидермолиза; болезни Гентингтона; болезни Краббе (инфантильной); ассоциированного с митохондриальной ДНК синдрома Ли и NARP; синдрома Леша-Найхана; ассоциированной с геном LIS1 лиссэнцефалии; синдрома Лоу; болезни "кленового сиропа"; синдрома дупликации MECP2; связанных с геном ATP7A нарушений обмена меди; связанной с геном LAMA2 мышечной дистрофии; недостаточности арилсульфатазы А; мукополисахаридоза I, II или III типов; связанных с биогенезом пероксисом нарушений, спектра заболеваний по типу синдрома Цельвегера; нарушений по типу нейродегенерации с накоплением железа в головном мозге; недостаточности кислой сфингомиелиназы; болезни Ниманна-Пика C типа; глициновой энцефалопатии; связанных с геном ARX нарушений; нарушений орнитинового цикла; связанного с геном COL1A1/2 несовершенного остеогенеза; синдромов удаления митохондриальной ДНК; связанных с геном PLP1 нарушений; синдрома Перри; синдрома Фелана-МакДермида; болезни накопления гликогена II типа (болезни Помпе) (инфантильной); связанных с геном MAPT нарушений; связанных с геном MECP2 нарушений; эпифизарной точечной хондродисплазии 1 типа костей верхних конечностей или бедренной кости; синдрома Робертса; болезни Сандхоффа; болезни Шиндлера - 1 типа; аденозиндезаминазной недостаточности; синдрома Смита-Лемли-Опитца; спинальной мышечной атрофии; спинально-церебеллярной атаксии с возникновением в младенческом возрасте; недостаточности гексозаминидазы А; танатофорной дисплазии 1 типа; связанных с геном коллагена VI типа нарушений; синдрома Ашера I типа; врожденной мышечной дистрофии; синдрома Вольфа-Хиршхорна; недостаточности лизосомной кислой липазы и пигментной ксеродермы.
Длительное введение белковых терапевтических средств может вызвать нежелательные иммунные ответы на данный белок. Иммуногенность белковых лекарственных средств может объясняться наличием нескольких иммунодоминантных эпитопов для T-лимфоцитов-хелперов (HTL). Путем снижения аффинности связывания этих эпитопов для HTL, содержащихся в данных белках, с MHC можно создавать лекарственные средства с более низкой иммуногенностью (Tangri S, et al. (“Rationally engineered therapeutic proteins with reduced immunogenicity” J Immunol. 2005 Mar 15;174(6):3187-96.) В настоящем изобретении иммуногенность фермента CRISPR, в частности, можно снизить, следуя подходу, впервые изложенному Tangri и соавт. в отношении эритропоэтина и впоследствии получившему развитие. Соответственно, для снижения иммуногенности фермента CRISPR (например, Cas9) у вида-хозяина (человека или другого вида) можно применять направленную эволюцию или рациональное проектирование.
У растений патогены часто являются специфичными по отношению к хозяину. Например, Fusarium oxysporum f. sp. lycopersici вызывает фузариозный вилт томата, но поражает только томат, а F. oxysporum f. dianthii и Puccinia graminis f. sp. tritici поражают только пшеницу. Растения обладают присущими и индуцированными защитными реакциями, обеспечивающими устойчивость к большинству патогенов. Мутации и события рекомбинации в поколениях растений приводят к генетической изменчивости, которая обуславливает восприимчивость, тем более, что патогены размножаются с большей частотой, чем растения. У растений может наблюдаться устойчивость видов-нехозяев, например, хозяин и патоген являются несовместимыми. Также может наблюдаться горизонтальная устойчивость, например, частичная устойчивость ко всем расам патогена, обычно контролируемая многими генами, и вертикальная устойчивость, например, полная устойчивость к некоторым расам патогена, но не к другим расам, обычно контролируемая несколькими генами. На уровне взаимодействия генов растения и патогены эволюционируют совместно, а генетические изменения одного уравновешивают изменения другого. Соответственно, используя естественную изменчивость, селекционеры комбинируют наиболее полезные гены для урожайности, качества, однородности, выносливости, устойчивости. Источники генов устойчивости включают нативные или чужеродные сорта, старинные сорта, родственные дикорастущие растения и индуцированные мутации, например, при обработке растительного материала мутагенными средствами. Применяя настоящее изобретение, селекционеры растений получают новый инструмент для индукции мутаций. Соответственно, специалист в данной области может проанализировать геном источников генов устойчивости, а в отношении сортов, имеющих желаемые характеристики или признаки, использовать настоящее изобретение для индукции появления генов устойчивости с большей точностью, чем в случае применявшихся ранее мутагенных средств, и, следовательно, ускорять и улучшать программы селекции растений.
Как будет понятно, предусматривается, что настоящую систему можно использовать для целенаправленного воздействия на любую представляющую интерес полинуклеотидную последовательность. Некоторые состояния или заболевания, которые можно эффективно лечить с использованием настоящей системы, включены в таблицы выше, и примеры известных на данный момент генов, ассоциированных с такими состояниями, также предоставлены в них. Тем не менее, гены, приведенные в качестве примеров, не являются исчерпывающими.
ПРИМЕРЫ
Следующие примеры приведены с целью иллюстрации различных вариантов осуществления настоящего изобретения и не предназначены для ограничения настоящего изобретения каким-либо образом. Данные примеры совместно со способами, описанными в данном документе, в настоящее время отражают предпочтительные варианты осуществления, являются иллюстративными и не предназначены для ограничения объема настоящего изобретения. Изменения в данном документе и другие применения, которые охватываются сутью настоящего изобретения, как определено объемом формулы изобретения, будут очевидны специалистам в данной области.
Пример 1: Активность комплекса CRISPR в ядре эукариотической клетки
Примером системы CRISPR II типа является локус CRISPR II типа из Streptococcus pyogenes SF370, который содержит кластер из 4 генов Cas9, Cas1, Cas2 и Csn1, а также два некодирующих элемента РНК, tracrRNA и характерный массив повторяющихся последовательностей (прямых повторов), чередующихся с короткими фрагментами неповторяющихся последовательностей (спейсерами, примерно 30 п.о. каждый). В этой системе двухцепочечный разрыв (DSB) целевой ДНК образовывается в ходе четырех последовательных стадий (фигура 2A). Во-первых, две некодирующие РНК, массив pre-crRNA и tracrRNA транскрибируются с локуса CRISPR. Во-вторых, tracrRNA гибридизируется с прямыми повторами pre-crRNA, которая затем процессируется в зрелые crRNA, содержащие индивидуальные спейсерные последовательности. В-третьих, комплекс зрелая crRNA:tracrRNA направляет Cas9 к ДНК-мишени, состоящей из протоспейсера и соответствующего PAM, посредством образования гетеродуплекса между спейсерным участком crRNA и протоспейсерной ДНК. И наконец, Cas9 опосредует расщепление целевой ДНК выше PAM с образованием DSB внутри протоспейсера (фигура 2A). В данном примере описывается иллюстративный способ для приспособления этой РНК-программируемой нуклеазной системы к управлению активностью комплекса CRISPR в ядрах эукариотических клеток.
Для улучшения экспрессии компонентов CRISPR в клетках млекопитающих два гена из локуса 1 SF370 Streptococcus pyogenes (S. pyogenes) были кодон-оптимизированы, Cas9 (SpCas9) и РНКаза III (SpRNase III). Для обеспечения ядерной локализации клеточный сигнал ядерной локализации (NLS) включали в амино(N-) или карбоксильные(C-) терминальные области и SpCas9, и SpRNase III (Фигура 2B). Для обеспечения визуализации экспрессии белков ген флуоресцентного белка в качестве маркера также включали в N- или C-терминальные области обоих белков (фигура 2B). Также был создан вариант SpCas9 с NLS, прикрепленным и к N-, и к C-терминальным областям (2xNLS-SpCas9). Конструкции, содержащие слитый с NLS SpCas9 и SpRNase III трансфицировали в клетки почки эмбриона человека (HEK) 293FT, и было обнаружено, что относительное положение NLS относительно SpCas9 и SpRNase III влияет на их эффективность ядерной локализации. Хотя C-терминального NLS было достаточно для нацеливания SpRNase III в ядро, прикрепление одной копии этих конкретных NLS либо к N-, либо к C-терминальной области SpCas9 не было способно обеспечить адекватную ядерную локализацию в этой системе. В этом примере C-терминальный NLS был из нуклеоплазмина (KRPAATKKAGQAKKKK), а C-терминальный NLS был из большого Т-антигена SV40 (PKKKRKV). Из тестируемых вариантов SpCas9 только 2xNLS-SpCas9 проявлял ядерную локализацию (фигура 2B).
tracrRNA из локуса CRISPR S. pyogenes SF370 содержал два сайта инициации транскрипции, дающие начало двум транскриптам из 89 нуклеотидов (нт) и 171 нт, которые затем подвергались процессингу в идентичные зрелые tracrRNA из 75 нт. Более короткие tracrRNA из 89 нт отбирали на предмет экспрессии в клетках млекопитающих (экспрессирующая конструкция изображена на фигуре 6, с функциональностью, как определено по результатам анализа с помощью Surveryor, показанным на фигуре 6B). Сайты инициации транскрипции обозначены как +1, а также указаны терминатор транскрипции и последовательность, гибридизирующаяся с зондом при нозерн-блоттинге. Экспрессию подвергнутой процессингу tracrRNA также подтверждали с помощью нозерн-блоттинга. На фигуре 7C показаны результаты анализа с помощью нозерн-блоттинга общей РНК, экстрагированной из клеток 293FT, трансфицированных экспрессирующими конструкциями U6, несущими длинную или короткую tracrRNA, а также SpCas9 и DR-EMX1(1)-DR. Левая и правая секции получены с клетками 293FT, трансфицированными без или с SpRNase III, соответственно. U6 являются показателем для контроля загрузки при блоттинге с зондом, нацеленным на малую ядерную РНК (snRNA) U6 человека. Трансфекция экспрессирующей конструкции с короткой tracrRNA приводит к избыточным уровням подвергшейся процессингу формы tracrRNA (~75 п.о.). Очень низкие количества длинных tracrRNA обнаруживали при нозерн-блоттинге.
Для стимуляции точной инициации транскрипции промотор U6 на основе РНК-полимеразы III выбирали для управления экспрессией tracrRNA (фигура 2C). Подобным образом, конструкцию на основе промотора U6 разрабатывали для экспрессии массива pre-crRNA, состоящего из одного спейсера, фланкированного двумя прямыми повторами (DR, также включены в выражение “парные tracr- последовательности”; фигура 2C). Исходный спейсер был разработан для целенаправленного воздействия на целевой сайт из 33 пар оснований (п.о.) (протоспейсер из 30 п.о., а также последовательность мотива CRISPR (PAM) из 3 п.о., соответствующая мотиву узнавания NGG у Cas9) в локусе EMX1 человека (фигура 2C), ключевом гене в развитии коры головного мозга.
Клетки HEK 293FT трансфицировали комбинациями компонентов CRISPR для того, чтобы определить, возможно ли при гетерологичной экспрессии системы CRISPR (SpCas9, SpRNase III, tracrRNA и pre-crRNA) в клетках млекопитающих достичь целенаправленного расщепления хромосом млекопитающего. Поскольку DSB в ядрах млекопитающих подвергаются частичной репарации с помощью пути негомологичного соединения концов (NHEJ), который приводит к образованию вставок/делеций, анализ с помощью Surveyor использовали для выявления возможной активности для расщепления в целевом локусе EMX1 (см., например, Guschin et al., 2010, Methods Mol Biol 649: 247). Котрансфекция всех четырех компонентов CRISPR была способна индуцировать расщепления в протоспейсере до 5,0% (см. фигуру 2D). Котрансфекция всех компонентов CRISPR, за исключением SpRNase III, также индуцировала образование вставок/делеций в протоспейсере на уровне до 4,7%, что указывало на то, что могут существовать эндогенные РНКазы млекопитающих, которые способны помогать созреванию crRNA, такие как, например, родственные ферменты Dicer и Drosha. Удаление любого из трех остальных компонентов ликвидировало активность системы CRISPR для расщепления генома (фигура 2D). Секвенирование по Сэнгеру ампликонов, содержащих целевой локус, подтверждало активность для расщепления: на 43 подвергшихся секвенированию клонов было обнаружено 5 мутированных аллелей (11,6%). В подобных экспериментах с использованием ряда направляющих последовательностей процентные значения содержания вставок/делеций составляли до 29% (см. фигуры 4-7, 12 и 13). Эти результаты определяют трехкомпонентную систему для эффективной опосредованной CRISPR модификации генома в клетках млекопитающих.
Для оптимизации эффективности расщепления авторы данной заявки также определяли, влияют ли различные изоформы tracrRNA на эффективность расщепления, и обнаружили, что в этой иллюстративной системе только короткая (89 п.о.) форма транскрипта была способна опосредовать расщепление локуса генома EMX1. На фигуре 9 представлен дополнительный анализ процессинга crRNA в клетках млекопитающих с помощью нозерн-блоттинга. На фигуре 9A показано схематическое изображение вектора экспрессии для одного спейсера, фланкированного двумя прямыми повторами (DR-EMX1(1)-DR). Спейсер из 30 п.о. нацеленный на протоспейсер 1 локуса EMX1 человека и последовательности прямых повторов показаны в последовательности внизу фигуры 9A. Линия указывает на участок, обратно комплементарную последовательность которого использовали для создания зондов для нозерн-блоттинга для выявления crRNA EMX1(1). На фигуре 9B показаны результаты анализа с помощью нозерн-блоттинга общей РНК, экстрагированной из клеток 293FT, трансфицированных экспрессирующими конструкциями U6, несущими DR-EMX1(1)-DR. Левая и правая секции получены с клетками 293FT, трансфицированными без или с SpRNase III, соответственно. DR-EMX1(1)-DR подвергался процессингу в зрелые crRNA только в присутствии SpCas9 и короткой tracrRNA и не зависел от присутствия SpRNase III. Зрелая crRNA, обнаруженная в общей РНК трансфицированных 293FT, имела длину ~33 п.о. и была короче, чем зрелая crRNA из S. pyogenes длиной 39-42 п.о. Данные результаты демонстрируют, что систему CRISPR можно перенести в эукариотические клетки и перепрограммировать для облегчения расщепления эндогенных целевых полинуклеотидов млекопитающих.
На фигуре 2 показана бактериальная система CRISPR, описанная в этом примере. На фигуре 2A показано схематическое изображение локуса 1 CRISPR из Streptococcus pyogenes SF370 и предполагаемый механизм опосредованного CRISPR расщепления ДНК с помощью этой системы. Зрелая crRNA, подвергшаяся процессингу из массива прямых повторов-спейсеров, направляет Cas9 к мишеням в геноме, состоящим из комплементарных протоспейсеров и мотива, смежного с протоспейсером (PAM). При спаривании оснований мишень-спейсер Cas9 опосредует двухцепочечный разрыв в целевой ДНК. На фигуре 2B показано конструирование Cas9 S. pyogenes (SpCas9) и РНКазы III (SpRNase III) с клеточными сигналами ядерной локализации (NLS) для обеспечения импорта в ядро млекопитающих. На фигуре 2C показана экспрессия SpCas9 и SpRNase III у млекопитающих, управляемая конститутивным промотором EF1a, и массива tracrRNA и pre-crRNA (DR-спейсер-DR), управляемая промотором U6 РНК-полимеразы 3 для стимуляции точной инициации и терминации транскрипции. Протоспейсер из локуса EMX1 человека с удовлетворительной последовательностью PAM использовали в качестве спейсера в массиве pre-crRNA. На фигуре 2D показан анализ с помощью нуклеазы Surveyor для опосредованных SpCas9 минорных вставок и делеций. SpCas9 экспрессировался с SpRNase III, tracrRNA и массивом pre-crRNA, несущим целевой спейсер для EMX1, и без таковых. На фигуре 2E показано схематическое изображение спаривания оснований между целевым локусом и нацеленной на EMX1 crRNA, а также иллюстративная хроматограмма, на которой показана микроделеция, смежная по отношению к сайту расщепления SpCas9. На фигуре 2F показаны мутированные аллели, идентифицированные в результате анализа секвенирования 43 клональных ампликонов, показывающие разнообразие микровставок и микроделеций. Штрихами указаны удаленные основания, а невыровненные или несовпадающие основания указывают на вставки или мутации. Масштабная метка = 10 мкм.
Для дальнейшего упрощения трехкомпонентной системы адаптировали химерную crRNA-tracrRNA гибридную структуру, в которой зрелую crRNA (содержащую направляющую последовательность) сливали с частичной tracrRNA при помощи структуры по типу стебель-петля для имитации естественного дуплекса crRNA:tracrRNA (фигура 3A).
Направляющие последовательности можно встроить между сайтами BbsI с использованием гибридизированных олигонуклеотидов. Протоспейсеры на смысловой и антисмысловой нитях указаны выше и ниже последовательностей ДНК, соответственно. Степень модификации для локусов PVALB человека и Th мыши достигали 6,3% и 0,75%, соответственно, демонстрируя широкую применимость системы CRISPR при модификации различных локусов у многих организмов. Хотя при использовании химерных конструкций расщепление обнаруживали только с одним из трех спейсеров для каждого локуса, все целевые последовательности расщеплялись с эффективностью получения вставок/делеций, достигающей 27%, при использовании схемы с коэкспрессируемой pre-crRNA (фигуры 4 и 5).
На фигуре 5 представлена дополнительная иллюстрация того, что SpCas9 можно перепрограммировать для целенаправленного воздействия на несколько локусов генома в клетках млекопитающих. На фигуре 5A представлено схематическое изображение локуса EMX1 человека, на котором показано положение пяти протоспейсеров, указанных с помощью подчеркнутых последовательностей. На фигуре 5B представлено схематическое изображение комплекса pre-crRNA/trcrRNA, на котором показана гибридизация между участком прямого повтора в pre-crRNA и tracrRNA (вверху), и схематическое изображение химерной структуры РНК, содержащей направляющую последовательность из 20 п.о. и парную tracr-последовательность и tracr-последовательность, состоящие из неполного прямого повтора и последовательностей tracrRNA, гибридизованных в "шпилечную" структуру (внизу). Результаты анализа с помощью Surveyor со сравнением эффективности опосредованного Cas9 расщепления в пяти протоспейсерах в локусе EMX1 человека показаны на фигуре 5C. Целенаправленное воздействие на каждый протоспейсер осуществляли либо с использованием подвергнутого процессингу комплекса pre-crRNA/tracrRNA (crRNA), либо с использованием химерной РНК (chiRNA).
Поскольку вторичная структура РНК может быть важной для межмолекулярных взаимодействий, алгоритм предсказания структуры на основе минимальной свободной энергии и ансамбля взвешенных структур по Больцману использовали для сравнения предполагаемой вторичной структуры всех направляющих последовательностей, используемых в эксперименте с целенаправленным воздействием на геном (фигура 3B) (см., например, Gruber et al., 2008, Nucleic Acids Research, 36: W70). Анализ выявил, что в большинстве случаев эффективные направляющие последовательности в контексте химерной crRNA, по сути, не содержали мотивов вторичной структуры, тогда как неэффективные направляющие последовательности с большей вероятностью образовывали внутренние вторичные структуры, которые могут препятствовать спариванию оснований с ДНК целевого протоспейсера. Следовательно, возможно, что вариабельность во вторичной структуре спейсера может оказывать воздействие на эффективность опосредованной CRISPR интерференции при использовании химерной crRNA.
На фигуре 3 показан пример векторов экспрессии. На фигуре 3A представлено схематическое изображение бицистронного вектора для управления экспрессией химерной синтетической конструкции crRNA-tracrRNA (химерной РНК), а также SpCas9. Химерная направляющая РНК содержит направляющую последовательность из 20 п.о., соответствующую протоспейсеру в геномном целевом сайте. На фигуре 3B представлено схематическое изображение, на котором показаны направляющие последовательности, нацеленные на локусы EMX1, PVALB человека и Th мыши, а также их предсказанные вторичные структуры. Эффективность модификации в каждом целевом сайте указана ниже рисунка вторичной структуры РНК (EMX1, n = 216 считываемых фрагментов при секвенировании ампликонов; PVALB, n = 224 считываемых фрагментов; Th, n = 265 считываемых фрагментов). Представлены результаты по алгоритму укладки каждого основания, окрашенного соответственно его возможности принятия предсказанной вторичной структуры, как указано с помощью цветной шкалы, воспроизведенной на фигуре 3B в виде серой шкалы. Структуры дополнительных векторов для SpCas9 показаны на фигуре 3A, в том числе отдельные векторы экспрессии, включающие промотор U6, сцепленный с сайтом встраивания для направляющего олигонуклеотида, и промотор Cbh, сцепленный с кодирующей последовательностью SpCas9.
Для того, чтобы определить, способны ли спейсеры с вторичными структурами функционировать в прокариотических клетках, где в естественных условиях функционируют CRISPR, интерференцию при трансформации плазмидами, несущими протоспейсеры, исследовали в штамме E. coli, гетерологично экспрессирующем локус 1 CRISPR S. pyogenes SF370 (фигура 3C). Локус CRISPR клонировали в низкокопийный вектор экспрессии E. coli и массив crRNA замещали одним спейсером, фланкированным парой DR (pCRISPR). Штаммы E. coli, несущие разные плазмиды pCRISPR, трансформировали контрольными плазмидами, содержащими соответствующие протоспейсер и последовательности PAM (фигура 3C). При анализе у бактерий все спейсеры способствовали эффективной CRISPR-интерференции (фигура 3C). Эти результаты указывают на то, что могут существовать дополнительные факторы, влияющие на эффективность активности CRISPR в клетках млекопитающих.
Для исследования специфичности опосредованного CRISPR расщепления эффект однонуклеотидных мутаций в направляющей последовательности в отношении расщепления протоспейсера в геноме млекопитающих анализировали с использованием ряда целенаправленно воздействующих на EMX химерных crRNA с единичными точковыми мутациями (фигура 4A). На фигуре 4B показаны результаты анализа с помощью нуклеазы Surveyor со сравнением эффективности расщепления Cas9 при спаривании с различными мутантными химерными РНК. Несовпадение одного основания в участке вплоть до 12 п.о. с 5' в PAM, по сути, прекращало расщепление генома SpCas9, тогда как спейсеры с мутациями в положениях, расположенных в более отдаленных положениях выше относительно хода транскрипции сохраняли активность в отношении исходного протоспейсера-мишени (фигура 4B). В дополнение к PAM, SpCas9 характеризуется специфичностью в отношении одного основания в последних 12 п.о. спейсера. Кроме того, CRISPR способен опосредовать расщепление генома столь же эффективно, как и пара нуклеаз TALE (TALEN), целенаправленно воздействующих на тот же протоспейсер EMX1. На фигуре 4C представлено схематическое изображение, на котором показана структура TALEN, целенаправленно воздействующих на EMX1, и на фигуре 4D показано сравнение эффективности TALEN и Cas9 (n=3) при разгонке в геле продуктов, полученных в результате анализа с помощью Surveyor.
Установив набор компонентов для достижения опосредованного CRISPR редактирования генов в клетках млекопитающих посредством склонного к ошибкам механизма NHEJ, исследовали способность CRISPR к стимуляции гомологичной рекомбинации (HR), высокоточный путь репарации генов для создания точных редакционных изменений в геноме. SpCas9 дикого типа способен опосредовать сайт-специфические DSB, которые могут репарироваться как с помощью NHEJ, так и HR. Кроме того, замену аспартата на аланин (D10A) в каталитическом домене RuvC I в SpCas9 производили посредством методик генетической инженерии для превращения нуклеазы в никазу (SpCas9n; проиллюстрировано на фигуре 5A) (см., например, Sapranausaks et al., 2011, Cucleic Acis Research, 39: 9275; Gasiunas et al., 2012, Proc. Natl. Acad. Sci. USA, 109:E2579) так, чтобы надрезанная геномная ДНК подвергалась высокоточной репарации с использованием гомологичной рекомбинации (HDR). Анализ с помощью Surveyor подтвердил, что SpCas9n не создает вставок/делеций в протоспейсере-мишени EMX1. Как показано на фигуре 5B, коэкспрессия целенаправленно воздействующей на EMX1 химерной crRNA с SpCas9 давала вставки/делеции в целевом сайте, тогда как коэкспрессия с SpCas9n - нет (n=3). Более того, при секвенировании 327 ампликонов не обнаружили каких-либо вставок/делеций, индуцированных SpCas9n. Для исследования опосредованной CRISPR HR при совместной трансфекции клеток HEK 293FT химерной РНК, целенаправленно воздействующей на EMX1, hSpCas9 или hSpCas9n, выбирали тот же локус, также как и матрицу для HR для введения пары сайтов рестрикции (HindIII и NheI) возле протоспейсера. На фигуре 5C приведена схематическая иллюстрация стратегии HR с относительными положениями точек рекомбинации и последовательностей для гибридизации праймеров (стрелки). SpCas9 и SpCas9n действительно катализировали интеграцию матрицы для HR в локус EMX1. ПЦР амплификация целевого участка с последующим рестрикционным расщеплением HindIII выявила продукты расщепления, соответствующие ожидаемым размерам фрагментов (стрелки на результатах анализа полиморфизма длин рестрикционных фрагментов с помощью гель-электрофореза, показанных на фигуре 5D), причем SpCas9 и SpCas9n опосредуют подобные уровни эффективности HR. Заявители дополнительно подтверждали HR с использованием секвенирования геномных ампликонов по Сэнгеру (фигура 5E). Эти результаты демонстрировали пригодность CRISPR для облегчения целенаправленной вставки генов в геном млекопитающего. С учетом специфичности к мишени в 14 п.о. (12 п.о. от спейсера и 2 п.о. от PAM) SpCas9 дикого типа доступность никазы может значительно снизить вероятность нецелевой модификации, поскольку одноцепочечные разрывы не являются субстратами для склонного к ошибкам пути NHEJ.
Экспрессирующие конструкции, имитирующие естественную архитектуру локусов CRISPR с собранными в массив спейсерами (фигура 2A), создавали для исследования возможности мультиплексного целенаправленного воздействия на последовательности. При использовании одного массива CRISPR, кодирующего пару спейсеров, нацеленных на EMX1 и PVALB, обнаруживали эффективное расщепление в обоих локусах (фигура 4F, на которой показаны как схематическая структура массива crRNA, так и блот, полученный после анализа с помощью Surveyor, показывающий эффективное опосредование расщепления). Также исследовали целенаправленную делецию геномных участков большего размера посредством одновременных DSB с использованием спейсеров против двух мишеней в EMX1, разделенных 119 п.о., и обнаруженная эффективность делеции составляла 1,6% (3 из 182 ампликонов; фигура 5G). Это демонстрирует, что система CRISPR может опосредовать мультиплексное редактирование в пределах одного генома.
Пример 2: модификации и альтернативы системы CRISPR
Возможность применения РНК для программирования специфичного к последовательности расщепления ДНК определяет новый класс инструментов для конструирования генома для разнообразных исследовательских и промышленных применений. Несколько аспектов системы CRISPR можно дополнительно улучшить для повышения эффективности и универсальности целенаправленного воздействия с помощью CRISPR. Оптимальная активность Cas9 может зависеть от доступности несвязанного Mg2+ на уровнях, которые превышают имеющиеся в ядре млекопитающего (см., например, Jinek et al., 2012, Science, 337:816), и предпочтение в отношении мотива NGG непосредственно ниже протоспейсера ограничивает способность к целенаправленному воздействию в среднем на каждые 12 п.о. в геноме человека. Некоторые из этих затруднений можно преодолеть путем изучения разнообразия локусов CRISPR в микробном метагеноме (см., например, Makarova et al., 2011, Nat Rev Microbiol, 9:467). Другие локусы CRISPR можно переместить в микроокружение клетки млекопитающего с помощью способа, подобного описанному в примере 1. Эффективность модификации в каждом целевом сайте указана ниже вторичных структур РНК. Алгоритм, генерирующий цвета структуры каждого основания соответственно его возможности принятия предсказанной вторичной структуры. РНК направляющих спейсеров 1 и 2 индуцировали 14% и 6,4%, соответственно. Статистический анализ активности для расщепления по биологическим копиям в этих двух протоспейсерных сайтах также приведен на фигуре 7.
Пример 3: алгоритм выбора примерной целевой последовательности
Создали компьютерную программу для идентификации кандидатных целевых последовательностей CRISPR на обеих нитях вводимой последовательности ДНК на основе длины желаемой направляющей последовательности и мотива последовательности CRISPR (PAM) для определенного фермента CRISPR. Например, целевые сайты для Cas9 из S. pyogenes с последовательностями PAM NGG можно идентифицировать путем поиска в отношении 5'-Nx-NGG-3' как на вводимой последовательности, так и на последовательности, обратно-комплементарной вводимой. Подобным образом, целевые сайты для Cas9 CRISPR1 S. thermophilus с последовательностью PAM NNAGAAW, можно идентифицировать путем поиска в отношении 5'-Nx-NNAGAAW-3' как на вводимой последовательности, так и на последовательности, обратно-комплементарной вводимой. Подобным образом, целевые сайты для Cas9 CRISPR3 S. thermophilus с последовательностью PAM NGGNG можно идентифицировать путем поиска в отношении 5'-Nx-NGGNG-3' как на вводимой последовательности, так и на последовательности, обратно-комплементарной вводимой. Значение “x” в Nx может фиксироваться программой или может быть определено пользователем, как, например, 20.
Поскольку несколько случаев появления целевого сайта ДНК в геноме могут приводить к неспецифическому редактированию генома, после идентификации всех возможных сайтов программа профильтровывает последовательности, исходя из количества раз, когда они встречаются в соответствующем эталонном геноме. Для тех ферментов CRISPR, для которых специфичность к последовательности определяется "затравочной" последовательностью, такой как находящаяся в 11-12 п.о. в направлении 5' от последовательности PAM, в том числе сама последовательность PAM, стадия фильтрования может основываться на "затравочной" последовательности. Следовательно, во избежание редактирования в дополнительных локусах генома результаты фильтруют, исходя из числа случаев обнаружения последовательности затравки:PAM в подходящем геноме. Пользователь может иметь возможность выбора длины затравочной последовательности. Пользователь также может иметь возможность определять число случаев обнаружения последовательности затравки:PAM в геноме применительно к прохождению фильтра. По умолчанию установлен скрининг в отношении уникальных последовательностей. Уровень фильтрования изменяют путем изменения как длины затравочной последовательности, так и числа случаев обнаружения последовательности в геноме. В качестве дополнения или альтернативы, программа может обеспечивать последовательность направляющей последовательности, комплементарную сообщенной(ым) целевой(ым) последовательности(ям) путем обеспечения последовательности, обратно комплементарной идентифицированной(ым) целевой(ым) последовательности(ям).
Дальнейшие детали способов и алгоритмов для оптимизации выбора последовательности можно найти в заявке на патент США с серийным номером TBA (общая ссылка BI-2012/084 44790.11.2022); включенной в данный документ при помощи ссылки.
Пример 4: оценка гибридов нескольких химерных crRNA-tracrRNA
В данном примере описаны результаты, полученные для химерных РНК (chiRNA; содержащие направляющую последовательность, парную tracr-последовательность и tracr-последовательность в одном транскрипте), имеющих tracr-последовательности, которые включают фрагменты последовательности tracrRNA дикого типа с разной длиной. На фигуре 18a показано схематическое изображение бицистронного вектора экспрессии для химерной РНК и Cas9. Cas9 управляется промотором CBh, а химерная РНК управляется промотором U6. Химерная направляющая РНК состоит из направляющей последовательности (Ns) из 20 п.о., соединенной с tracr-последовательностью (проходящей от первого “U” в нижней нити к концу транскрипта), которая усечена в разных указанных положениях. Направляющие и tracr-последовательности разделены парной tracr-последовательностью GUUUUAGAGCUA, за которой следует последовательность петли GAAA. Результаты анализов с помощью SURVEYOR в отношении опосредованных Cas9 вставок/делеций в локусах EMX1 и PVALB человека показаны на фигурах 18b и 18c, соответственно. Стрелки указывают на ожидаемые фрагменты, полученные в результате расщепления с помощью SURVEYOR. ChiRNA показаны путем обозначения их “+n”, а crRNA относится к гибридной РНК, в которой направляющие и tracr-последовательности экспрессируются в виде раздельных транскриптов. Количественный анализ этих результатов, выполненный в трех повторностях, проиллюстрирован с помощью гистограмм на фигурах 11a и 11b, соответствующих фигурам 10b и 10c, соответственно (“N.D.” означает отсутствие обнаруженных вставок/делеций). ID (идентификационные данные) протоспейсеров и их соответствующей мишени в геноме, последовательность протоспейсера, последовательность PAM и положение нити приведены в таблице D. Направляющие последовательности разработаны так, чтобы они были комплементарны полной последовательности протоспейсера в случае отдельных транскриптов в гибридной системе или только подчеркнутой части в случае химерных РНК.
Таблица D:
протоспейсера
в геноме
Клеточная культура и трансфекция
Линию клеток почки эмбриона человека (HEK) 293FT (Life Technologies) поддерживали в среде Игла в модификации Дульбекко (DMEM), дополненной 10% фетальной бычьей сыворотки (HyClone), 2 мМ GlutaMAX (Life Technologies), 100 ЕД/мл пенициллина и 100 мкг/мл стрептомицина, при 37°C с инкубированием при 5% CO CO2. Клетки 293FT засевали в 24-луночные планшеты (Corning) за 24 часа до трансфекции при плотности 150000 клеток на лунку. Клетки трансфицировали с применением Lipofectamine 2000 (Life Technologies), следуя рекомендованному производителем протоколу. Для каждой лунки 24-луночного планшета использовали в общей сложности 500 нг плазмид.
Анализ с помощью SURVEYOR на предмет наличия модификации генома
Клетки 293FT трансфицировали плазмидной ДНК, как описано выше. Клетки инкубировали при 37°C в течение 72 часов после трансфекции перед экстракцией геномной ДНК. Геномную ДНК экстрагировали с помощью раствора QuickExtract DNA Extraction Solution (Epicentre), следуя протоколу производителя. Вкратце, осажденные центрифугированием клетки ресуспендировали в растворе QuickExtract solution и инкубировали при 65°C в течение 15 минут и 98°C в течение 10 минут. Геномный участок, фланкирующий целевой сайт CRISPR каждого гена, амплифицировали с помощью ПЦР (праймеры перечислены в таблице E) и продукты очищали с применением колонки QiaQuick Spin Column (Qiagen), следуя протоколу производителя. В общей сложности 400 нг очищенных ПЦР-продуктов смешивали с 2 мкл 10X ПЦР-буфера для ДНК-полимеразы Taq (Enzymatics) и воды сверхвысокой чистоты до конечного объема 20 мкл и подвергали процессу повторного отжига для обеспечения образования гетеродуплекса: 95°C в течение 10 мин., линейное снижение температуры с 95°C до 85°C со скоростью 2°C/с, с 85°C до 25°C со скоростью 0,25°C/с и с выдерживанием при 25°C в течение 1 минуты. После повторного отжига продукты обрабатывали нуклеазой SURVEYOR и энхансером S SURVEYOR (Transgenomics), следуя рекомендованному производителем протоколу, и анализировали в 4-20% полиакриламидных гелях Novex TBE (Life Technologies). Гели окрашивали красителем ДНК SYBR Gold (Life Technologies) в течение 30 минут и получали изображение с помощью системы обработки изображений Gel Doc gel imaging system (Bio-rad). Количественный анализ основывался на относительных интенсивностях полос.
Таблица E:
Вычислительная идентификация уникальных целевых сайтов CRISPR
Для идентификации уникальных целевых сайтов для фермента Cas9 (SpCas9) S. pyogenes SF370 в геноме человека, мыши, крысы, данио, плодовой мухи и C. elegans был разработан пакет программ для сканирования обеих нитей последовательности ДНК и идентификации всех возможных целевых сайтов SpCas9. Для этого примера каждый целевой сайт SpCas9 был оперативно определен как последовательность из 20 п.о., за которой следует последовательность мотива, смежного с протоспейсером, (PAM) NGG, при этом были определены все последовательности, удовлетворяющие определению 5'-N20-NGG-3' на всех хромосомах. Для предотвращения неспецифического редактирования генома после идентификации всех потенциальных сайтов все целевые сайты фильтровали, исходя из количества раз, когда они встречаются в соответствующем эталонном геноме. Для того чтобы извлечь пользу из специфичности к последовательности активности Cas9, обеспечиваемой "затравочной" последовательностью, которой может быть, например, последовательность из приблизительно 11-12 п.о. 5' от последовательности PAM, при этом последовательности 5'-NNNNNNNNNN-NGG-3' выбирали как уникальные в соответствующем геноме. Все геномные последовательности загружали из геномного браузера UCSC (геном человека hg19, геном мыши mm9, геном крысы rn5, геном данио danRer7, геном D. melanogaster dm4 и геном C. elegans ce10). Все результаты поиска доступны для просмотра с использованием информации из геномного браузера UCSC. Иллюстративная визуализация некоторых целевых сайтов в геноме человека представлена на фигуре 22.
Первоначально целенаправленному воздействию подвергали три сайта в пределах локуса EMX1 в клетках HEK 293FT человека. Эффективность модификации генома каждой chiРНК оценивали с использованием анализа с помощью нуклеазы SURVEYOR, который позволяет обнаруживать мутации, возникающие в результате двухцепочечных разрывов (DSB) ДНК и их последующей репарации с помощью пути репарации повреждения ДНК за счет негомологичного соединения концов (NHEJ). В конструкциях, обозначенных chiRNA(+n), указывается, что нуклеотиды в количестве до +n нуклеотида tracrRNA дикого типа включены в химерную РНК-конструкцию, при этом для n используются значения 48, 54, 67 и 85. Химерные РНК, содержащие более длинные фрагменты tracrRNA дикого типа (chiRNA(+67) и chiRNA(+85)), опосредовали расщепление ДНК во всех трех целевых сайтах EMX1, причем chiRNA(+85), в частности, демонстрировал значительно более высокие уровни расщепления ДНК, чем соответствующие гибриды crRNA/tracrRNA, у которых направляющие и tracr-последовательности экспрессируются в отдельных транскриптах (фигуры 10b и 10a). Два сайта в локусе PVALB, которые не давали обнаруживаемого расщепления с использованием гибридной системы (направляющая последовательность и tracr-последовательность, экспрессируемые в виде отдельных транскриптов), также подвергались целенаправленному воздействию с использованием chiRNA. сhiRNA(+67) и chiRNA(+85) были способны опосредовать значительное расщепление в двух протоспейсерах в PVALB (фигуры 10c и 10b).
Для всех пяти мишеней в локусах EMX1 и PVALB наблюдали соответствующее повышение эффективности модификации генома с увеличением длины tracr-последовательности. Не вдаваясь в какую-либо теорию, вторичная структура, формируемая 3' концом tracrRNA, может играть роль в увеличении скорости образования комплекса CRISPR. Иллюстрация предсказанных вторичных структур для каждой химерной РНК, использованной в этом примере, представлена на фигуре 21. Вторичную структуру предсказывали с применением RNAfold (http://RNA.tbi.univie.ac.at/cgi-bin/RNAfold.cgi) с использованием минимальной свободной энергии и алгоритма функции распределения. Псевдоцвет для каждого основания (воспроизведен в серой шкале) указывает на возможность спаривания. По причине того, что chiRNA с более длинными tracr-последовательностями были способны расщеплять мишени, которые не были расщеплены нативными гибридами crRNA/tracrRNA CRISPR, возможно, что химерная РНК может загружаться на Cas9 более эффективно, чем ее нативный гибридный аналог. Для обеспечения применения Cas9 для сайт-специфического редактирования генома в эукариотических клетках и организмах все предсказанные уникальные целевые сайты для Cas9 S. pyogenes определяли путем вычислений в геномах человека, мыши, крысы, данио, C. elegans и D. melanogaster. Химерные РНК можно разрабатывать для ферментов Cas9 из других микроорганизмов для расширения целевого пространства CRISPR РНК-программируемых нуклеаз.
На фигурах 11 и 21 показаны примерные бицистронные векторы экспрессии для экспрессии химерной РНК, включающие tracr-последовательность РНК дикого типа вплоть до нуклеотида +85 и SpCas9 с последовательностями ядерной локализации SpCas9 экспрессируется с промотора CBh и терминируется polyA-сигналом bGH (bGH pA). Расширенная последовательность, показанная непосредственно под схематическим изображением, соответствует участку, окружающему направляющую последовательность сайта встраивания, и включает от 5' дo 3', 3'-часть промотора U6 (первый заштрихованный участок), сайты расщепления BbsI (стрелки), неполный прямой повтор (парная tracr-последовательность GTTTTAGAGCTA, подчеркнутая), последовательность петли GAAA и +85 tracr-последовательность (подчеркнутая последовательность, следующая за последовательностью петли). Иллюстративное встраивание направляющей последовательности изображено ниже сайта встраивания направляющей последовательности, при этом нуклеотиды направляющей последовательности для выбранной мишени представлены как “N”.
Последовательности, описанные в приведенных выше примерах, представляют собой следующие (полинуклеотидные последовательности представлены от 5' к 3').
U6 с короткой tracrRNA (Streptococcus pyogenes SF370):
GAGGGCCTATTTCCCATGATTCCTTCATATTTGCATATACGATACAAGGCTGTTAGAGAGATAATTGGAATTAATTTGACTGTAAACACAAAGATATTAGTACAAAATACGTGACGTAGAAAGTAATAATTTCTTGGGTAGTTTGCAGTTTTAAAATTATGTTTTAAAATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCGATTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCGGAACCATTCAAAACAGCATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTT (жирный шрифт = последовательность tracrRNA; подчеркивание = терминаторная последовательность).
U6 с длинной tracrRNA (Streptococcus pyogenes SF370):
GAGGGCCTATTTCCCATGATTCCTTCATATTTGCATATACGATACAAGGCTGTTAGAGAGATAATTGGAATTAATTTGACTGTAAACACAAAGATATTAGTACAAAATACGTGACGTAGAAAGTAATAATTTCTTGGGTAGTTTGCAGTTTTAAAATTATGTTTTAAAATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCGATTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCGGTAGTATTAAGTATTGTTTTATGGCTGATAAATTTCTTTGAATTTCTCCTTGATTATTTGTTATAAAAGTTATAAAATAATCTTGTTGGAACCATTCAAAACAGCATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTT.
U6-DR-BbsI-остов-DR (Streptococcus pyogenes SF370):
GAGGGCCTATTTCCCATGATTCCTTCATATTTGCATATACGATACAAGGCTGTTAGAGAGATAATTGGAATTAATTTGACTGTAAACACAAAGATATTAGTACAAAATACGTGACGTAGAAAGTAATAATTTCTTGGGTAGTTTGCAGTTTTAAAATTATGTTTTAAAATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCGATTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCGGGTTTTAGAGCTATGCTGTTTTGAATGGTCCCAAAACGGGTCTTCGAGAAGACGTTTTAGAGCTATGCTGTTTTGAATGGTCCCAAAAC.
U6-химерная РНК-BbsI-остов (Streptococcus pyogenes SF370)
GAGGGCCTATTTCCCATGATTCCTTCATATTTGCATATACGATACAAGGCTGTTAGAGAGATAATTGGAATTAATTTGACTGTAAACACAAAGATATTAGTACAAAATACGTGACGTAGAAAGTAATAATTTCTTGGGTAGTTTGCAGTTTTAAAATTATGTTTTAAAATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCGATTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCGGGTCTTCGAGAAGACCTGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCG.
NLS-SpCas9-EGFP:
MDYKDHDGDYKDHDIDYKDDDDKMAPKKKRKVGIHGVPAADKKYSIGLDIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDHIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGDAAAVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYK.
SpCas9-EGFP-NLS:
MDKKYSIGLDIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDHIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGDAAAVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYKKRPAATKKAGQAKKKK.
NLS-SpCas9-EGFP-NLS:
MDYKDHDGDYKDHDIDYKDDDDKMAPKKKRKVGIHGVPAADKKYSIGLDIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDHIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGDAAAVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTYGVQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYKKRPAATKKAGQAKKKK.
NLS-SpCas9-NLS:
MDYKDHDGDYKDHDIDYKDDDDKMAPKKKRKVGIHGVPAADKKYSIGLDIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDHIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGDKRPAATKKAGQAKKKK.
NLS-mCherry-SpRNase3:
MFLFLSLTSFLSSSRTLVSKGEEDNMAIIKEFMRFKVHMEGSVNGHEFEIEGEGEGRPYEGTQTAKLKVTKGGPLPFAWDILSPQFMYGSKAYVKHPADIPDYLKLSFPEGFKWERVMNFEDGGVVTVTQDSSLQDGEFIYKVKLRGTNFPSDGPVMQKKTMGWEASSERMYPEDGALKGEIKQRLKLKDGGHYDAEVKTTYKAKKPVQLPGAYNVNIKLDITSHNEDYTIVEQYERAEGRHSTGGMDELYKGSKQLEELLSTSFDIQFNDLTLLETAFTHTSYANEHRLLNVSHNERLEFLGDAVLQLIISEYLFAKYPKKTEGDMSKLRSMIVREESLAGFSRFCSFDAYIKLGKGEEKSGGRRRDTILGDLFEAFLGALLLDKGIDAVRRFLKQVMIPQVEKGNFERVKDYKTCLQEFLQTKGDVAIDYQVISEKGPAHAKQFEVSIVVNGAVLSKGLGKSKKLAEQDAAKNALAQLSEV.
SpRNase3-mCherry-NLS:
MKQLEELLSTSFDIQFNDLTLLETAFTHTSYANEHRLLNVSHNERLEFLGDAVLQLIISEYLFAKYPKKTEGDMSKLRSMIVREESLAGFSRFCSFDAYIKLGKGEEKSGGRRRDTILGDLFEAFLGALLLDKGIDAVRRFLKQVMIPQVEKGNFERVKDYKTCLQEFLQTKGDVAIDYQVISEKGPAHAKQFEVSIVVNGAVLSKGLGKSKKLAEQDAAKNALAQLSEVGSVSKGEEDNMAIIKEFMRFKVHMEGSVNGHEFEIEGEGEGRPYEGTQTAKLKVTKGGPLPFAWDILSPQFMYGSKAYVKHPADIPDYLKLSFPEGFKWERVMNFEDGGVVTVTQDSSLQDGEFIYKVKLRGTNFPSDGPVMQKKTMGWEASSERMYPEDGALKGEIKQRLKLKDGGHYDAEVKTTYKAKKPVQLPGAYNVNIKLDITSHNEDYTIVEQYERAEGRHSTGGMDELYKKRPAATKKAGQAKKKK.
NLS-SpCas9n-NLS (D10A мутация никазы представлена в нижнем регистре):
MDYKDHDGDYKDHDIDYKDDDDKMAPKKKRKVGIHGVPAADKKYSIGLaIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEATRLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVDEVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFIQLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGLTPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNTEITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEFYKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLKDNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMTNFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRKVTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIVLTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDFLKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVVDELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDHIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGDKRPAATKKAGQAKKKK.
hEMX1-HR-матрица-HindII-NheI:
GAATGCTGCCCTCAGACCCGCTTCCTCCCTGTCCTTGTCTGTCCAAGGAGAATGAGGTCTCACTGGTGGATTTCGGACTACCCTGAGGAGCTGGCACCTGAGGGACAAGGCCCCCCACCTGCCCAGCTCCAGCCTCTGATGAGGGGTGGGAGAGAGCTACATGAGGTTGCTAAGAAAGCCTCCCCTGAAGGAGACCACACAGTGTGTGAGGTTGGAGTCTCTAGCAGCGGGTTCTGTGCCCCCAGGGATAGTCTGGCTGTCCAGGCACTGCTCTTGATATAAACACCACCTCCTAGTTATGAAACCATGCCCATTCTGCCTCTCTGTATGGAAAAGAGCATGGGGCTGGCCCGTGGGGTGGTGTCCACTTTAGGCCCTGTGGGAGATCATGGGAACCCACGCAGTGGGTCATAGGCTCTCTCATTTACTACTCACATCCACTCTGTGAAGAAGCGATTATGATCTCTCCTCTAGAAACTCGTAGAGTCCCATGTCTGCCGGCTTCCAGAGCCTGCACTCCTCCACCTTGGCTTGGCTTTGCTGGGGCTAGAGGAGCTAGGATGCACAGCAGCTCTGTGACCCTTTGTTTGAGAGGAACAGGAAAACCACCCTTCTCTCTGGCCCACTGTGTCCTCTTCCTGCCCTGCCATCCCCTTCTGTGAATGTTAGACCCATGGGAGCAGCTGGTCAGAGGGGACCCCGGCCTGGGGCCCCTAACCCTATGTAGCCTCAGTCTTCCCATCAGGCTCTCAGCTCAGCCTGAGTGTTGAGGCCCCAGTGGCTGCTCTGGGGGCCTCCTGAGTTTCTCATCTGTGCCCCTCCCTCCCTGGCCCAGGTGAAGGTGTGGTTCCAGAACCGGAGGACAAAGTACAAACGGCAGAAGCTGGAGGAGGAAGGGCCTGAGTCCGAGCAGAAGAAGAAGGGCTCCCATCACATCAACCGGTGGCGCATTGCCACGAAGCAGGCCAATGGGGAGGACATCGATGTCACCTCCAATGACaagcttgctagcGGTGGGCAACCACAAACCCACGAGGGCAGAGTGCTGCTTGCTGCTGGCCAGGCCCCTGCGTGGGCCCAAGCTGGACTCTGGCCACTCCCTGGCCAGGCTTTGGGGAGGCCTGGAGTCATGGCCCCACAGGGCTTGAAGCCCGGGGCCGCCATTGACAGAGGGACAAGCAATGGGCTGGCTGAGGCCTGGGACCACTTGGCCTTCTCCTCGGAGAGCCTGCCTGCCTGGGCGGGCCCGCCCGCCACCGCAGCCTCCCAGCTGCTCTCCGTGTCTCCAATCTCCCTTTTGTTTTGATGCATTTCTGTTTTAATTTATTTTCCAGGCACCACTGTAGTTTAGTGATCCCCAGTGTCCCCCTTCCCTATGGGAATAATAAAAGTCTCTCTCTTAATGACACGGGCATCCAGCTCCAGCCCCAGAGCCTGGGGTGGTAGATTCCGGCTCTGAGGGCCAGTGGGGGCTGGTAGAGCAAACGCGTTCAGGGCCTGGGAGCCTGGGGTGGGGTACTGGTGGAGGGGGTCAAGGGTAATTCATTAACTCCTCTCTTTTGTTGGGGGACCCTGGTCTCTACCTCCAGCTCCACAGCAGGAGAAACAGGCTAGACATAGGGAAGGGCCATCCTGTATCTTGAGGGAGGACAGGCCCAGGTCTTTCTTAACGTATTGAGAGGTGGGAATCAGGCCCAGGTAGTTCAATGGGAGAGGGAGAGTGCTTCCCTCTGCCTAGAGACTCTGGTGGCTTCTCCAGTTGAGGAGAAACCAGAGGAAAGGGGAGGATTGGGGTCTGGGGGAGGGAACACCATTCACAAAGGCTGACGGTTCCAGTCCGAAGTCGTGGGCCCACCAGGATGCTCACCTGTCCTTGGAGAACCGCTGGGCAGGTTGAGACTGCAGAGACAGGGCTTAAGGCTGAGCCTGCAACCAGTCCCCAGTGACTCAGGGCCTCCTCAGCCCAAGAAAGAGCAACGTGCCAGGGCCCGCTGAGCTCTTGTGTTCACCTG.
NLS-StCsn1-NLS:
MKRPAATKKAGQAKKKKSDLVLGLDIGIGSVGVGILNKVTGEIIHKNSRIFPAAQAENNLVRRTNRQGRRLARRKKHRRVRLNRLFEESGLITDFTKISINLNPYQLRVKGLTDELSNEELFIALKNMVKHRGISYLDDASDDGNSSVGDYAQIVKENSKQLETKTPGQIQLERYQTYGQLRGDFTVEKDGKKHRLINVFPTSAYRSEALRILQTQQEFNPQITDEFINRYLEILTGKRKYYHGPGNEKSRTDYGRYRTSGETLDNIFGILIGKCTFYPDEFRAAKASYTAQEFNLLNDLNNLTVPTETKKLSKEQKNQIINYVKNEKAMGPAKLFKYIAKLLSCDVADIKGYRIDKSGKAEIHTFEAYRKMKTLETLDIEQMDRETLDKLAYVLTLNTEREGIQEALEHEFADGSFSQKQVDELVQFRKANSSIFGKGWHNFSVKLMMELIPELYETSEEQMTILTRLGKQKTTSSSNKTKYIDEKLLTEEIYNPVVAKSVRQAIKIVNAAIKEYGDFDNIVIEMARETNEDDEKKAIQKIQKANKDEKDAAMLKAANQYNGKAELPHSVFHGHKQLATKIRLWHQQGERCLYTGKTISIHDLINNSNQFEVDHILPLSITFDDSLANKVLVYATANQEKGQRTPYQALDSMDDAWSFRELKAFVRESKTLSNKKKEYLLTEEDISKFDVRKKFIERNLVDTRYASRVVLNALQEHFRAHKIDTKVSVVRGQFTSQLRRHWGIEKTRDTYHHHAVDALIIAASSQLNLWKKQKNTLVSYSEDQLLDIETGELISDDEYKESVFKAPYQHFVDTLKSKEFEDSILFSYQVDSKFNRKISDATIYATRQAKVGKDKADETYVLGKIKDIYTQDGYDAFMKIYKKDKSKFLMYRHDPQTFEKVIEPILENYPNKQINEKGKEVPCNPFLKYKEEHGYIRKYSKKGNGPEIKSLKYYDSKLGNHIDITPKDSNNKVVLQSVSPWRADVYFNKTTGKYEILGLKYADLQFEKGTGTYKISQEKYNDIKKKEGVDSDSEFKFTLYKNDLLLVKDTETKEQQLFRFLSRTMPKQKHYVELKPYDKQKFEGGEALIKVLGNVANSGQCKKGLGKSNISIYKVRTDVLGNQHIIKNEGDKPKLDFKRPAATKKAGQAKKKK.
U6-St_tracrRNA(7-97):
GAGGGCCTATTTCCCATGATTCCTTCATATTTGCATATACGATACAAGGCTGTTAGAGAGATAATTGGAATTAATTTGACTGTAAACACAAAGATATTAGTACAAAATACGTGACGTAGAAAGTAATAATTTCTTGGGTAGTTTGCAGTTTTAAAATTATGTTTTAAAATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCGATTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCGTTACTTAAATCTTGCAGAAGCTACAAAGATAAGGCTTCATGCCGAAATCAACACCCTGTCATTTTATGGCAGGGTGTTTTCGTTATTTAA.
U6-DR-спейсер-DR (S. pyogenes SF370)
gagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccgggttttagagctatgctgttttgaatggtcccaaaacNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNgttttagagctatgctgttttgaatggtcccaaaacTTTTTTT (нижний регистр, подчеркивание = прямой повтор; N = направляющая последовательность; жирный шрифт = терминатор).
Химерная РНК, содержащая +48 tracr RNA (S. pyogenes SF370)
gagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccNNNNNNNNNNNNNNNNNNNNgttttagagctagaaatagcaagttaaaataaggctagtccgTTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Химерная РНК, содержащая +54 tracr RNA (S. pyogenes SF370)
gagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccNNNNNNNNNNNNNNNNNNNNgttttagagctagaaatagcaagttaaaataaggctagtccgttatcaTTTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Химерная РНК, содержащая +67 tracr RNA (S. pyogenes SF370)
gagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccNNNNNNNNNNNNNNNNNNNNgttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagtgTTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор)
Химерная РНК, содержащая +85 tracr RNA (S. pyogenes SF370)
gagggcctatttcccatgattccttcatatttgcatatacgatacaaggctgttagagagataattggaattaatttgactgtaaacacaaagatattagtacaaaatacgtgacgtagaaagtaataatttcttgggtagtttgcagttttaaaattatgttttaaaatggactatcatatgcttaccgtaacttgaaagtatttcgatttcttggctttatatatcttgtggaaaggacgaaacaccNNNNNNNNNNNNNNNNNNNNgttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgcTTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
CBh-NLS-SpCas9-NLS
CGTTACATAACTTACGGTAAATGGCCCGCCTGGCTGACCGCCCAACGACCCCCGCCCATTGACGTCAATAATGACGTATGTTCCCATAGTAACGCCAATAGGGACTTTCCATTGACGTCAATGGGTGGAGTATTTACGGTAAACTGCCCACTTGGCAGTACATCAAGTGTATCATATGCCAAGTACGCCCCCTATTGACGTCAATGACGGTAAATGGCCCGCCTGGCATTATGCCCAGTACATGACCTTATGGGACTTTCCTACTTGGCAGTACATCTACGTATTAGTCATCGCTATTACCATGGTCGAGGTGAGCCCCACGTTCTGCTTCACTCTCCCCATCTCCCCCCCCTCCCCACCCCCAATTTTGTATTTATTTATTTTTTAATTATTTTGTGCAGCGATGGGGGCGGGGGGGGGGGGGGGGCGCGCGCCAGGCGGGGCGGGGCGGGGCGAGGGGCGGGGCGGGGCGAGGCGGAGAGGTGCGGCGGCAGCCAATCAGAGCGGCGCGCTCCGAAAGTTTCCTTTTATGGCGAGGCGGCGGCGGCGGCGGCCCTATAAAAAGCGAAGCGCGCGGCGGGCGGGAGTCGCTGCGACGCTGCCTTCGCCCCGTGCCCCGCTCCGCCGCCGCCTCGCGCCGCCCGCCCCGGCTCTGACTGACCGCGTTACTCCCACAGGTGAGCGGGCGGGACGGCCCTTCTCCTCCGGGCTGTAATTAGCTGAGCAAGAGGTAAGGGTTTAAGGGATGGTTGGTTGGTGGGGTATTAATGTTTAATTACCTGGAGCACCTGCCTGAAATCACTTTTTTTCAGGTTGGaccggtgccaccATGGACTATAAGGACCACGACGGAGACTACAAGGATCATGATATTGATTACAAAGACGATGACGATAAGATGGCCCCAAAGAAGAAGCGGAAGGTCGGTATCCACGGAGTCCCAGCAGCCGACAAGAAGTACAGCATCGGCCTGGACATCGGCACCAACTCTGTGGGCTGGGCCGTGATCACCGACGAGTACAAGGTGCCCAGCAAGAAATTCAAGGTGCTGGGCAACACCGACCGGCACAGCATCAAGAAGAACCTGATCGGAGCCCTGCTGTTCGACAGCGGCGAAACAGCCGAGGCCACCCGGCTGAAGAGAACCGCCAGAAGAAGATACACCAGACGGAAGAACCGGATCTGCTATCTGCAAGAGATCTTCAGCAACGAGATGGCCAAGGTGGACGACAGCTTCTTCCACAGACTGGAAGAGTCCTTCCTGGTGGAAGAGGATAAGAAGCACGAGCGGCACCCCATCTTCGGCAACATCGTGGACGAGGTGGCCTACCACGAGAAGTACCCCACCATCTACCACCTGAGAAAGAAACTGGTGGACAGCACCGACAAGGCCGACCTGCGGCTGATCTATCTGGCCCTGGCCCACATGATCAAGTTCCGGGGCCACTTCCTGATCGAGGGCGACCTGAACCCCGACAACAGCGACGTGGACAAGCTGTTCATCCAGCTGGTGCAGACCTACAACCAGCTGTTCGAGGAAAACCCCATCAACGCCAGCGGCGTGGACGCCAAGGCCATCCTGTCTGCCAGACTGAGCAAGAGCAGACGGCTGGAAAATCTGATCGCCCAGCTGCCCGGCGAGAAGAAGAATGGCCTGTTCGGCAACCTGATTGCCCTGAGCCTGGGCCTGACCCCCAACTTCAAGAGCAACTTCGACCTGGCCGAGGATGCCAAACTGCAGCTGAGCAAGGACACCTACGACGACGACCTGGACAACCTGCTGGCCCAGATCGGCGACCAGTACGCCGACCTGTTTCTGGCCGCCAAGAACCTGTCCGACGCCATCCTGCTGAGCGACATCCTGAGAGTGAACACCGAGATCACCAAGGCCCCCCTGAGCGCCTCTATGATCAAGAGATACGACGAGCACCACCAGGACCTGACCCTGCTGAAAGCTCTCGTGCGGCAGCAGCTGCCTGAGAAGTACAAAGAGATTTTCTTCGACCAGAGCAAGAACGGCTACGCCGGCTACATTGACGGCGGAGCCAGCCAGGAAGAGTTCTACAAGTTCATCAAGCCCATCCTGGAAAAGATGGACGGCACCGAGGAACTGCTCGTGAAGCTGAACAGAGAGGACCTGCTGCGGAAGCAGCGGACCTTCGACAACGGCAGCATCCCCCACCAGATCCACCTGGGAGAGCTGCACGCCATTCTGCGGCGGCAGGAAGATTTTTACCCATTCCTGAAGGACAACCGGGAAAAGATCGAGAAGATCCTGACCTTCCGCATCCCCTACTACGTGGGCCCTCTGGCCAGGGGAAACAGCAGATTCGCCTGGATGACCAGAAAGAGCGAGGAAACCATCACCCCCTGGAACTTCGAGGAAGTGGTGGACAAGGGCGCTTCCGCCCAGAGCTTCATCGAGCGGATGACCAACTTCGATAAGAACCTGCCCAACGAGAAGGTGCTGCCCAAGCACAGCCTGCTGTACGAGTACTTCACCGTGTATAACGAGCTGACCAAAGTGAAATACGTGACCGAGGGAATGAGAAAGCCCGCCTTCCTGAGCGGCGAGCAGAAAAAGGCCATCGTGGACCTGCTGTTCAAGACCAACCGGAAAGTGACCGTGAAGCAGCTGAAAGAGGACTACTTCAAGAAAATCGAGTGCTTCGACTCCGTGGAAATCTCCGGCGTGGAAGATCGGTTCAACGCCTCCCTGGGCACATACCACGATCTGCTGAAAATTATCAAGGACAAGGACTTCCTGGACAATGAGGAAAACGAGGACATTCTGGAAGATATCGTGCTGACCCTGACACTGTTTGAGGACAGAGAGATGATCGAGGAACGGCTGAAAACCTATGCCCACCTGTTCGACGACAAAGTGATGAAGCAGCTGAAGCGGCGGAGATACACCGGCTGGGGCAGGCTGAGCCGGAAGCTGATCAACGGCATCCGGGACAAGCAGTCCGGCAAGACAATCCTGGATTTCCTGAAGTCCGACGGCTTCGCCAACAGAAACTTCATGCAGCTGATCCACGACGACAGCCTGACCTTTAAAGAGGACATCCAGAAAGCCCAGGTGTCCGGCCAGGGCGATAGCCTGCACGAGCACATTGCCAATCTGGCCGGCAGCCCCGCCATTAAGAAGGGCATCCTGCAGACAGTGAAGGTGGTGGACGAGCTCGTGAAAGTGATGGGCCGGCACAAGCCCGAGAACATCGTGATCGAAATGGCCAGAGAGAACCAGACCACCCAGAAGGGACAGAAGAACAGCCGCGAGAGAATGAAGCGGATCGAAGAGGGCATCAAAGAGCTGGGCAGCCAGATCCTGAAAGAACACCCCGTGGAAAACACCCAGCTGCAGAACGAGAAGCTGTACCTGTACTACCTGCAGAATGGGCGGGATATGTACGTGGACCAGGAACTGGACATCAACCGGCTGTCCGACTACGATGTGGACCATATCGTGCCTCAGAGCTTTCTGAAGGACGACTCCATCGACAACAAGGTGCTGACCAGAAGCGACAAGAACCGGGGCAAGAGCGACAACGTGCCCTCCGAAGAGGTCGTGAAGAAGATGAAGAACTACTGGCGGCAGCTGCTGAACGCCAAGCTGATTACCCAGAGAAAGTTCGACAATCTGACCAAGGCCGAGAGAGGCGGCCTGAGCGAACTGGATAAGGCCGGCTTCATCAAGAGACAGCTGGTGGAAACCCGGCAGATCACAAAGCACGTGGCACAGATCCTGGACTCCCGGATGAACACTAAGTACGACGAGAATGACAAGCTGATCCGGGAAGTGAAAGTGATCACCCTGAAGTCCAAGCTGGTGTCCGATTTCCGGAAGGATTTCCAGTTTTACAAAGTGCGCGAGATCAACAACTACCACCACGCCCACGACGCCTACCTGAACGCCGTCGTGGGAACCGCCCTGATCAAAAAGTACCCTAAGCTGGAAAGCGAGTTCGTGTACGGCGACTACAAGGTGTACGACGTGCGGAAGATGATCGCCAAGAGCGAGCAGGAAATCGGCAAGGCTACCGCCAAGTACTTCTTCTACAGCAACATCATGAACTTTTTCAAGACCGAGATTACCCTGGCCAACGGCGAGATCCGGAAGCGGCCTCTGATCGAGACAAACGGCGAAACCGGGGAGATCGTGTGGGATAAGGGCCGGGATTTTGCCACCGTGCGGAAAGTGCTGAGCATGCCCCAAGTGAATATCGTGAAAAAGACCGAGGTGCAGACAGGCGGCTTCAGCAAAGAGTCTATCCTGCCCAAGAGGAACAGCGATAAGCTGATCGCCAGAAAGAAGGACTGGGACCCTAAGAAGTACGGCGGCTTCGACAGCCCCACCGTGGCCTATTCTGTGCTGGTGGTGGCCAAAGTGGAAAAGGGCAAGTCCAAGAAACTGAAGAGTGTGAAAGAGCTGCTGGGGATCACCATCATGGAAAGAAGCAGCTTCGAGAAGAATCCCATCGACTTTCTGGAAGCCAAGGGCTACAAAGAAGTGAAAAAGGACCTGATCATCAAGCTGCCTAAGTACTCCCTGTTCGAGCTGGAAAACGGCCGGAAGAGAATGCTGGCCTCTGCCGGCGAACTGCAGAAGGGAAACGAACTGGCCCTGCCCTCCAAATATGTGAACTTCCTGTACCTGGCCAGCCACTATGAGAAGCTGAAGGGCTCCCCCGAGGATAATGAGCAGAAACAGCTGTTTGTGGAACAGCACAAGCACTACCTGGACGAGATCATCGAGCAGATCAGCGAGTTCTCCAAGAGAGTGATCCTGGCCGACGCTAATCTGGACAAAGTGCTGTCCGCCTACAACAAGCACCGGGATAAGCCCATCAGAGAGCAGGCCGAGAATATCATCCACCTGTTTACCCTGACCAATCTGGGAGCCCCTGCCGCCTTCAAGTACTTTGACACCACCATCGACCGGAAGAGGTACACCAGCACCAAAGAGGTGCTGGACGCCACCCTGATCCACCAGAGCATCACCGGCCTGTACGAGACACGGATCGACCTGTCTCAGCTGGGAGGCGACTTTCTTTTTCTTAGCTTGACCAGCTTTCTTAGTAGCAGCAGGACGCTTTAA (подчеркивание = NLS-hSpCas9-NLS).
Иллюстративная химерная РНК для Cas9 из CRISPR1 LMD-9 S. thermophilus (с PAM NNAGAAW)
NNNNNNNNNNNNNNNNNNNNgtttttgtactctcaagatttaGAAAtaaatcttgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgttttcgttatttaaTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Иллюстративная химерная РНК для Cas9 из CRISPR1 LMD-9 S. thermophilus (с PAM NNAGAAW)
NNNNNNNNNNNNNNNNNNNNgtttttgtactctcaGAAAtgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgttttcgttatttaaTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Иллюстративная химерная РНК для Cas9 из CRISPR1 LMD-9 S. thermophilus (с PAM NNAGAAW)
NNNNNNNNNNNNNNNNNNNNgtttttgtactctcaGAAAtgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgtTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Иллюстративная химерная РНК для Cas9 из CRISPR1 LMD-9 S. thermophilus (с PAM NNAGAAW)
NNNNNNNNNNNNNNNNNNNNgttattgtactctcaagatttaGAAAtaaatcttgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgttttcgttatttaaTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Иллюстративная химерная РНК для Cas9 из CRISPR1 LMD-9 S. thermophilus (с PAM NNAGAAW)
NNNNNNNNNNNNNNNNNNNNgttattgtactctcaGAAAtgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgttttcgttatttaaTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Иллюстративная химерная РНК для Cas9 из CRISPR1 LMD-9 S. thermophilus (с PAM NNAGAAW)
NNNNNNNNNNNNNNNNNNNNgttattgtactctcaGAAAtgcagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgtTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Иллюстративная химерная РНК для Cas9 из CRISPR1 LMD-9 S. thermophilus (с PAM NNAGAAW)
NNNNNNNNNNNNNNNNNNNNgttattgtactctcaagatttaGAAAtaaatcttgcagaagctacaatgataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgttttcgttatttaaTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Иллюстративная химерная РНК для Cas9 из CRISPR1 LMD-9 S. thermophilus (с PAM NNAGAAW)
NNNNNNNNNNNNNNNNNNNNgttattgtactctcaGAAAtgcagaagctacaatgataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgttttcgttatttaaTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Иллюстративная химерная РНК для Cas9 из CRISPR1 LMD-9 S. thermophilus (с PAM NNAGAAW)
NNNNNNNNNNNNNNNNNNNNgttattgtactctcaGAAAtgcagaagctacaatgataaggcttcatgccgaaatcaacaccctgtcattttatggcagggtgtTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Иллюстративная химерная РНК для Cas9 из CRISPR1 LMD-9 S. thermophilus (с PAM NGGNG)
NNNNNNNNNNNNNNNNNNNNgttttagagctgtgGAAAcacagcgagttaaaataaggcttagtccgtactcaacttgaaaaggtggcaccgattcggtgtTTTTTT (N = направляющая последовательность; первое подчеркивание = парная tracr-последовательность; второе подчеркивание = tracr-последовательность; жирный шрифт = терминатор).
Кодон-оптимизированный вариант Cas9 из локуса CRISPR3 LMD-9 S. thermophilus (с NLS и на 5'-, и на 3'-концах)
ATGAAAAGGCCGGCGGCCACGAAAAAGGCCGGCCAGGCAAAAAAGAAAAAGACCAAGCCCTACAGCATCGGCCTGGACATCGGCACCAATAGCGTGGGCTGGGCCGTGACCACCGACAACTACAAGGTGCCCAGCAAGAAAATGAAGGTGCTGGGCAACACCTCCAAGAAGTACATCAAGAAAAACCTGCTGGGCGTGCTGCTGTTCGACAGCGGCATTACAGCCGAGGGCAGACGGCTGAAGAGAACCGCCAGACGGCGGTACACCCGGCGGAGAAACAGAATCCTGTATCTGCAAGAGATCTTCAGCACCGAGATGGCTACCCTGGACGACGCCTTCTTCCAGCGGCTGGACGACAGCTTCCTGGTGCCCGACGACAAGCGGGACAGCAAGTACCCCATCTTCGGCAACCTGGTGGAAGAGAAGGCCTACCACGACGAGTTCCCCACCATCTACCACCTGAGAAAGTACCTGGCCGACAGCACCAAGAAGGCCGACCTGAGACTGGTGTATCTGGCCCTGGCCCACATGATCAAGTACCGGGGCCACTTCCTGATCGAGGGCGAGTTCAACAGCAAGAACAACGACATCCAGAAGAACTTCCAGGACTTCCTGGACACCTACAACGCCATCTTCGAGAGCGACCTGTCCCTGGAAAACAGCAAGCAGCTGGAAGAGATCGTGAAGGACAAGATCAGCAAGCTGGAAAAGAAGGACCGCATCCTGAAGCTGTTCCCCGGCGAGAAGAACAGCGGAATCTTCAGCGAGTTTCTGAAGCTGATCGTGGGCAACCAGGCCGACTTCAGAAAGTGCTTCAACCTGGACGAGAAAGCCAGCCTGCACTTCAGCAAAGAGAGCTACGACGAGGACCTGGAAACCCTGCTGGGATATATCGGCGACGACTACAGCGACGTGTTCCTGAAGGCCAAGAAGCTGTACGACGCTATCCTGCTGAGCGGCTTCCTGACCGTGACCGACAACGAGACAGAGGCCCCACTGAGCAGCGCCATGATTAAGCGGTACAACGAGCACAAAGAGGATCTGGCTCTGCTGAAAGAGTACATCCGGAACATCAGCCTGAAAACCTACAATGAGGTGTTCAAGGACGACACCAAGAACGGCTACGCCGGCTACATCGACGGCAAGACCAACCAGGAAGATTTCTATGTGTACCTGAAGAAGCTGCTGGCCGAGTTCGAGGGGGCCGACTACTTTCTGGAAAAAATCGACCGCGAGGATTTCCTGCGGAAGCAGCGGACCTTCGACAACGGCAGCATCCCCTACCAGATCCATCTGCAGGAAATGCGGGCCATCCTGGACAAGCAGGCCAAGTTCTACCCATTCCTGGCCAAGAACAAAGAGCGGATCGAGAAGATCCTGACCTTCCGCATCCCTTACTACGTGGGCCCCCTGGCCAGAGGCAACAGCGATTTTGCCTGGTCCATCCGGAAGCGCAATGAGAAGATCACCCCCTGGAACTTCGAGGACGTGATCGACAAAGAGTCCAGCGCCGAGGCCTTCATCAACCGGATGACCAGCTTCGACCTGTACCTGCCCGAGGAAAAGGTGCTGCCCAAGCACAGCCTGCTGTACGAGACATTCAATGTGTATAACGAGCTGACCAAAGTGCGGTTTATCGCCGAGTCTATGCGGGACTACCAGTTCCTGGACTCCAAGCAGAAAAAGGACATCGTGCGGCTGTACTTCAAGGACAAGCGGAAAGTGACCGATAAGGACATCATCGAGTACCTGCACGCCATCTACGGCTACGATGGCATCGAGCTGAAGGGCATCGAGAAGCAGTTCAACTCCAGCCTGAGCACATACCACGACCTGCTGAACATTATCAACGACAAAGAATTTCTGGACGACTCCAGCAACGAGGCCATCATCGAAGAGATCATCCACACCCTGACCATCTTTGAGGACCGCGAGATGATCAAGCAGCGGCTGAGCAAGTTCGAGAACATCTTCGACAAGAGCGTGCTGAAAAAGCTGAGCAGACGGCACTACACCGGCTGGGGCAAGCTGAGCGCCAAGCTGATCAACGGCATCCGGGACGAGAAGTCCGGCAACACAATCCTGGACTACCTGATCGACGACGGCATCAGCAACCGGAACTTCATGCAGCTGATCCACGACGACGCCCTGAGCTTCAAGAAGAAGATCCAGAAGGCCCAGATCATCGGGGACGAGGACAAGGGCAACATCAAAGAAGTCGTGAAGTCCCTGCCCGGCAGCCCCGCCATCAAGAAGGGAATCCTGCAGAGCATCAAGATCGTGGACGAGCTCGTGAAAGTGATGGGCGGCAGAAAGCCCGAGAGCATCGTGGTGGAAATGGCTAGAGAGAACCAGTACACCAATCAGGGCAAGAGCAACAGCCAGCAGAGACTGAAGAGACTGGAAAAGTCCCTGAAAGAGCTGGGCAGCAAGATTCTGAAAGAGAATATCCCTGCCAAGCTGTCCAAGATCGACAACAACGCCCTGCAGAACGACCGGCTGTACCTGTACTACCTGCAGAATGGCAAGGACATGTATACAGGCGACGACCTGGATATCGACCGCCTGAGCAACTACGACATCGACCATATTATCCCCCAGGCCTTCCTGAAAGACAACAGCATTGACAACAAAGTGCTGGTGTCCTCCGCCAGCAACCGCGGCAAGTCCGATGATGTGCCCAGCCTGGAAGTCGTGAAAAAGAGAAAGACCTTCTGGTATCAGCTGCTGAAAAGCAAGCTGATTAGCCAGAGGAAGTTCGACAACCTGACCAAGGCCGAGAGAGGCGGCCTGAGCCCTGAAGATAAGGCCGGCTTCATCCAGAGACAGCTGGTGGAAACCCGGCAGATCACCAAGCACGTGGCCAGACTGCTGGATGAGAAGTTTAACAACAAGAAGGACGAGAACAACCGGGCCGTGCGGACCGTGAAGATCATCACCCTGAAGTCCACCCTGGTGTCCCAGTTCCGGAAGGACTTCGAGCTGTATAAAGTGCGCGAGATCAATGACTTTCACCACGCCCACGACGCCTACCTGAATGCCGTGGTGGCTTCCGCCCTGCTGAAGAAGTACCCTAAGCTGGAACCCGAGTTCGTGTACGGCGACTACCCCAAGTACAACTCCTTCAGAGAGCGGAAGTCCGCCACCGAGAAGGTGTACTTCTACTCCAACATCATGAATATCTTTAAGAAGTCCATCTCCCTGGCCGATGGCAGAGTGATCGAGCGGCCCCTGATCGAAGTGAACGAAGAGACAGGCGAGAGCGTGTGGAACAAAGAAAGCGACCTGGCCACCGTGCGGCGGGTGCTGAGTTATCCTCAAGTGAATGTCGTGAAGAAGGTGGAAGAACAGAACCACGGCCTGGATCGGGGCAAGCCCAAGGGCCTGTTCAACGCCAACCTGTCCAGCAAGCCTAAGCCCAACTCCAACGAGAATCTCGTGGGGGCCAAAGAGTACCTGGACCCTAAGAAGTACGGCGGATACGCCGGCATCTCCAATAGCTTCACCGTGCTCGTGAAGGGCACAATCGAGAAGGGCGCTAAGAAAAAGATCACAAACGTGCTGGAATTTCAGGGGATCTCTATCCTGGACCGGATCAACTACCGGAAGGATAAGCTGAACTTTCTGCTGGAAAAAGGCTACAAGGACATTGAGCTGATTATCGAGCTGCCTAAGTACTCCCTGTTCGAACTGAGCGACGGCTCCAGACGGATGCTGGCCTCCATCCTGTCCACCAACAACAAGCGGGGCGAGATCCACAAGGGAAACCAGATCTTCCTGAGCCAGAAATTTGTGAAACTGCTGTACCACGCCAAGCGGATCTCCAACACCATCAATGAGAACCACCGGAAATACGTGGAAAACCACAAGAAAGAGTTTGAGGAACTGTTCTACTACATCCTGGAGTTCAACGAGAACTATGTGGGAGCCAAGAAGAACGGCAAACTGCTGAACTCCGCCTTCCAGAGCTGGCAGAACCACAGCATCGACGAGCTGTGCAGCTCCTTCATCGGCCCTACCGGCAGCGAGCGGAAGGGACTGTTTGAGCTGACCTCCAGAGGCTCTGCCGCCGACTTTGAGTTCCTGGGAGTGAAGATCCCCCGGTACAGAGACTACACCCCCTCTAGTCTGCTGAAGGACGCCACCCTGATCCACCAGAGCGTGACCGGCCTGTACGAAACCCGGATCGACCTGGCTAAGCTGGGCGAGGGAAAGCGTCCTGCTGCTACTAAGAAAGCTGGTCAAGCTAAGAAAAAGAAATAA.
Пример 5: оптимизация направляющей РНК для Cas9 Streptococcus pyogenes (называемого SpCas9)
Авторы данной заявки вносили мутации в tracrRNA и последовательности прямых повторов или вносили мутации в химерную направляющую РНК для повышения экспрессии РНК в клетках.
Оптимизация основана на наблюдении, что присутствовали фрагменты тимина (Ts) в tracrRNA и направляющей РНК, которые могли приводить к ранней терминации транскрипции посредством промотора pol 3. Таким образом заявители создавали следующие оптимизированные последовательности. Оптимизированная tracrRNA и соответствующий оптимизированный прямой повтор представлены в парах.
Оптимизированная tracrRNA 1 (мутация подчеркнута):
GGAACCATTCAtAACAGCATAGCAAGTTAtAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTT.
Оптимизированный прямой повтор 1 (мутация подчеркнута):
GTTaTAGAGCTATGCTGTTaTGAATGGTCCCAAAAC.
Оптимизированная tracrRNA 2 (мутация подчеркнута):
GGAACCATTCAAtACAGCATAGCAAGTTAAtATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTT.
Оптимизированный прямой повтор 2 (мутация подчеркнута):
GTaTTAGAGCTATGCTGTaTTGAATGGTCCCAAAAC.
Авторы данной заявки также оптимизировали химерную направляющую РНК для оптимальной активности в эукариотических клетках.
Исходная направляющая РНК:
NNNNNNNNNNNNNNNNNNNNGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTT.
Оптимизированная химерная направляющая последовательность РНК 1:
NNNNNNNNNNNNNNNNNNNNGTATTAGAGCTAGAAATAGCAAGTTAATATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTT.
Оптимизированная химерная направляющая последовательность РНК 2:
NNNNNNNNNNNNNNNNNNNNGTTTTAGAGCTATGCTGTTTTGGAAACAAAACAGCATAGCAAGTTAAAATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTT.
Оптимизированная химерная направляющая последовательность РНК 3:
NNNNNNNNNNNNNNNNNNNNGTATTAGAGCTATGCTGTATTGGAAACAATACAGCATAGCAAGTTAATATAAGGCTAGTCCGTTATCAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTTTTT.
Авторы данной заявки показали, что оптимизированная химерная направляющая РНК работает лучше, как показано на фигуре 9. Эксперимент проводили путем котрансфекции клеток 293FT Cas9 и ДНК-кассетой с U6-направляющей РНК для экспрессии одной из четырех форм РНК, показанных выше. Мишень направляющей РНК является таким же целевым сайтом в локусе EMX1 человека: “GTCACCTCCAATGACTAGGG”.
Пример 6: оптимизация Cas9 из CRISPR1 LMD-9 Streptococcus thermophilus (называемого St1Cas9)
Авторы данной заявки разрабатывали направляющие химерные РНК, как показано на фигуре 12.
Направляющие РНК St1Cas9 можно подвергать такому же типу оптимизации, как и направляющие РНК SpCas9, путем разрушения политиминовых фрагментов (Ts).
Пример 7: улучшение системы Cas9 для применения in vivo
Авторы данной заявки проводили поиск с использованием метагеномного подхода в отношении Cas9 с малым молекулярным весом. Большинство гомологов Cas9 являются достаточно большими. Например, SpCas9 имеет длину около 1368 а.к., что слишком много для легкой упаковки в вирусные векторы для доставки. Некоторые из последовательностей могли быть неверно аннотированы, и, таким образом, точная частота каждой длины не обязательно может быть достоверной. Тем не менее она дает некоторое представление о распределении белков Cas9 и указывает на то, что существуют более короткие гомологи Cas9.
С помощью анализа на основе расчетов авторы данной заявки обнаружили, что у штамма бактерии Campylobacter присутствуют два белка Cas9 с менее чем 1000 аминокислот. Последовательность для одного Cas9 из Campylobacter jejuni представлена ниже. При такой длине CjCas9 может быть легко упакован в AAV, лентивирусы, аденовирусы и другие вирусные векторы для надежной доставки в первичные клетки и in vivo в животные модели.
>Cas9 Campylobacter jejuni (CjCas9)
MARILAFDIGISSIGWAFSENDELKDCGVRIFTKVENPKTGESLALPRRLARSARKRLARRKARLNHLKHLIANEFKLNYEDYQSFDESLAKAYKGSLISPYELRFRALNELLSKQDFARVILHIAKRRGYDDIKNSDDKEKGAILKAIKQNEEKLANYQSVGEYLYKEYFQKFKENSKEFTNVRNKKESYERCIAQSFLKDELKLIFKKQREFGFSFSKKFEEEVLSVAFYKRALKDFSHLVGNCSFFTDEKRAPKNSPLAFMFVALTRIINLLNNLKNTEGILYTKDDLNALLNEVLKNGTLTYKQTKKLLGLSDDYEFKGEKGTYFIEFKKYKEFIKALGEHNLSQDDLNEIAKDITLIKDEIKLKKALAKYDLNQNQIDSLSKLEFKDHLNISFKALKLVTPLMLEGKKYDEACNELNLKVAINEDKKDFLPAFNETYYKDEVTNPVVLRAIKEYRKVLNALLKKYGKVHKINIELAREVGKNHSQRAKIEKEQNENYKAKKDAELECEKLGLKINSKNILKLRLFKEQKEFCAYSGEKIKISDLQDEKMLEIDHIYPYSRSFDDSYMNKVLVFTKQNQEKLNQTPFEAFGNDSAKWQKIEVLAKNLPTKKQKRILDKNYKDKEQKNFKDRNLNDTRYIARLVLNYTKDYLDFLPLSDDENTKLNDTQKGSKVHVEAKSGMLTSALRHTWGFSAKDRNNHLHHAIDAVIIAYANNSIVKAFSDFKKEQESNSAELYAKKISELDYKNKRKFFEPFSGFRQKVLDKIDEIFVSKPERKKPSGALHEETFRKEEEFYQSYGGKEGVLKALELGKIRKVNGKIVKNGDMFRVDIFKHKKTNKFYAVPIYTMDFALKVLPNKAVARSKKGEIKDWILMDENYEFCFSLYKDSLILIQTKDMQEPEFVYYNAFTSSTVSLIVSKHDNKFETLSKNQKILFKNANEKEVIAKSIGIQNLKVFEKYIVSALGEVTKAEFRQREDFKK.
Предполагаемый элемент tracrRNA для данного CjCas9 представляет собой:
TATAATCTCATAAGAAATTTAAAAAGGGACTAAAATAAAGAGTTTGCGGGACTCTGCGGGGTTACAATCCCCTAAAACCGCTTTTAAAATT.
Последовательность прямого повтора представляет собой:
ATTTTACCATAAAGAAATTTAAAAAGGGACTAAAAC.
Совместно свернутая структура tracrRNA и прямой повтор представлены на фигуре 6.
Пример химерной направляющей РНК для CjCas9 представляет собой:
NNNNNNNNNNNNNNNNNNNNGUUUUAGUCCCGAAAGGGACUAAAAUAAAGAGUUUGCGGGACUCUGCGGGGUUACAAUCCCCUAAAACCGCUUUU.
Авторы данной заявки также оптимизировали направляющую РНК для Cas9 с применением способов in vitro. На фигуре 18 показаны данные, полученные в результате in vitro оптимизации химерной направляющей РНК St1Cas9.
Несмотря на то, что предпочтительные варианты осуществления настоящего изобретения были показаны и описаны в данном документе, для специалиста в данной области будет очевидно, что такие варианты осуществления предоставлены только в качестве примера. Многочисленные вариации, изменения и замены теперь будут очевидны для специалиста в данной области без отступления от сути настоящего изобретения. Следует понимать, что различные альтернативные варианты вариантов осуществления настоящего изобретения, раскрытые в данном документе, можно применять при практическом осуществлении настоящего изобретения. Предполагают, что следующая формула изобретения определяет объем настоящего изобретения, и что, таким образом, охвачены способы и структуры в пределах объема данной формулы изобретения и их эквиваленты.
Пример 8: оптимизация Sa sgRNA
Авторы данной заявки разрабатывали пять вариантов sgRNA для SaCas9 для оптимальной усеченной архитектуры с наивысшей эффективностью расщепления. Кроме того, дуплексную систему нативный прямой повтор:tracr исследовали совместно с sgRNA. Гиды с указанными длинами совместно трансфицировали SaCas9 и исследовали в клетках HEK 293FT в отношении активности. Посредством в общей сложности 100 нг sgRNA U6-ПЦР-ампликона (или 50 нг прямого повтора и 50 нг tracrRNA) и 400 нг плазмид с SaCas9 совместно трансфицировали 200000 гепатоцитов мыши Hepa1-6 и ДНК собирали через 72 часа после трансфекции для анализа при помощи SURVEYOR. Результаты показаны на фиг. 23.
Библиографические ссылки
1. Urnov, F.D., Rebar, E.J., Holmes, M.C., Zhang, H.S. & Gregory, P.D. Genome editing with engineered zinc finger nucleases. Nat. Rev. Genet. 11, 636-646 (2010).
2. Bogdanove, A.J. & Voytas, D.F. TAL effectors: customizable proteins for DNA targeting. Science 333, 1843-1846 (2011).
3. Stoddard, B.L. Homing endonuclease structure and function. Q. Rev. Biophys. 38, 49-95 (2005).
4. Bae, T. & Schneewind, O. Allelic replacement in Staphylococcus aureus with inducible counter-selection. Plasmid 55, 58-63 (2006).
5. Sung, C.K., Li, H., Claverys, J.P. & Morrison, D.A. An rpsL cassette, janus, for gene replacement through negative selection in Streptococcus pneumoniae. Appl. Environ. Microbiol. 67, 5190-5196 (2001).
6. Sharan, S.K., Thomason, L.C., Kuznetsov, S.G. & Court, D.L. Recombineering: a homologous recombination-based method of genetic engineering. Nat. Protoc. 4, 206-223 (2009).
7. Jinek, M. et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816-821 (2012).
8. Deveau, H., Garneau, J.E. & Moineau, S. CRISPR-Cas system and its role in phage-bacteria interactions. Annu. Rev. Microbiol. 64, 475-493 (2010).
9. Horvath, P. & Barrangou, R. CRISPR-Cas, the immune system of bacteria and archaea. Science 327, 167-170 (2010).
10. Terns, M.P. & Terns, R.M. CRISPR-based adaptive immune systems. Curr. Opin. Microbiol. 14, 321-327 (2011).
11. van der Oost, J., Jore, M.M., Westra, E.R., Lundgren, M. & Brouns, S.J. CRISPR-based adaptive and heritable immunity in prokaryotes. Trends. Biochem. Sci. 34, 401-407 (2009).
12. Brouns, S.J. et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science 321, 960-964 (2008).
13. Carte, J., Wang, R., Li, H., Terns, R.M. & Terns, M.P. Cas6 is an endoribonuclease that generates guide RNAs for invader defense in prokaryotes. Genes Dev. 22, 3489-3496 (2008).
14. Deltcheva, E. et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature 471, 602-607 (2011).
15. Hatoum-Aslan, A., Maniv, I. & Marraffini, L.A. Mature clustered, regularly interspaced, short palindromic repeats RNA (crRNA) length is measured by a ruler mechanism anchored at the precursor processing site. Proc. Natl. Acad. Sci. U.S.A. 108, 21218-21222 (2011).
16. Haurwitz, R.E., Jinek, M., Wiedenheft, B., Zhou, K. & Doudna, J.A. Sequence- and structure-specific RNA processing by a CRISPR endonuclease. Science 329, 1355-1358 (2010).
17. Deveau, H. et al. Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. J. Bacteriol. 190, 1390-1400 (2008).
18. Gasiunas, G., Barrangou, R., Horvath, P. & Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc. Natl. Acad. Sci. U.S.A. (2012).
19. Makarova, K.S., Aravind, L., Wolf, Y.I. & Koonin, E.V. Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems. Biol. Direct. 6, 38 (2011).
20. Barrangou, R. RNA-mediated programmable DNA cleavage. Nat. Biotechnol. 30, 836-838 (2012).
21. Brouns, S.J. Molecular biology. A Swiss army knife of immunity. Science 337, 808-809 (2012).
22. Carroll, D. A CRISPR Approach to Gene Targeting. Mol. Ther. 20, 1658-1660 (2012).
23. Bikard, D., Hatoum-Aslan, A., Mucida, D. & Marraffini, L.A. CRISPR interference can prevent natural transformation and virulence acquisition during in vivo bacterial infection. Cell Host Microbe 12, 177-186 (2012).
24. Sapranauskas, R. et al. The Streptococcus thermophilus CRISPR-Cas system provides immunity in Escherichia coli. Nucleic Acids Res. (2011).
25. Semenova, E. et al. Interference by clustered regularly interspaced short palindromic repeat (CRISPR) RNA is governed by a seed sequence. Proc. Natl. Acad. Sci. U.S.A. (2011).
26. Wiedenheft, B. et al. RNA-guided complex from a bacterial immune system enhances target recognition through seed sequence interactions. Proc. Natl. Acad. Sci. U.S.A. (2011).
27. Zahner, D. & Hakenbeck, R. The Streptococcus pneumoniae beta-galactosidase is a surface protein. J. Bacteriol. 182, 5919-5921 (2000).
28. Marraffini, L.A., Dedent, A.C. & Schneewind, O. Sortases and the art of anchoring proteins to the envelopes of gram-positive bacteria. Microbiol. Mol. Biol. Rev. 70, 192-221 (2006).
29. Motamedi, M.R., Szigety, S.K. & Rosenberg, S.M. Double-strand-break repair recombination in Escherichia coli: physical evidence for a DNA replication mechanism in vivo. Genes Dev. 13, 2889-2903 (1999).
30. Hosaka, T. et al. The novel mutation K87E in ribosomal protein S12 enhances protein synthesis activity during the late growth phase in Escherichia coli. Mol. Genet. Genomics 271, 317-324 (2004).
31. Costantino, N. & Court, D.L. Enhanced levels of lambda Red-mediated recombinants in mismatch repair mutants. Proc. Natl. Acad. Sci. U.S.A. 100, 15748-15753 (2003).
32. Edgar, R. & Qimron, U. The Escherichia coli CRISPR system protects from lambda lysogenization, lysogens, and prophage induction. J. Bacteriol. 192, 6291-6294 (2010).
33. Marraffini, L.A. & Sontheimer, E.J. Self versus non-self discrimination during CRISPR RNA-directed immunity. Nature 463, 568-571 (2010).
34. Fischer, S. et al. An archaeal immune system can detect multiple Protospacer Adjacent Motifs (PAMs) to target invader DNA. J. Biol. Chem. 287, 33351-33363 (2012).
35. Gudbergsdottir, S. et al. Dynamic properties of the Sulfolobus CRISPR-Cas and CRISPR/Cmr systems when challenged with vector-borne viral and plasmid genes and protospacers. Mol. Microbiol. 79, 35-49 (2011).
36. Wang, H.H. et al. Genome-scale promoter engineering by coselection MAGE. Nat Methods 9, 591-593 (2012).
37. Cong, L. et al. Multiplex Genome Engineering Using CRISPR-Cas Systems. Science в печати (2013).
38. Mali, P. et al. RNA-Guided Human Genome Engineering via Cas9. Science в печати (2013).
39. Hoskins, J. et al. Genome of the bacterium Streptococcus pneumoniae strain R6. J. Bacteriol. 183, 5709-5717 (2001).
40. Havarstein, L.S., Coomaraswamy, G. & Morrison, D.A. An unmodified heptadecapeptide pheromone induces competence for genetic transformation in Streptococcus pneumoniae. Proc. Natl. Acad. Sci. U.S.A. 92, 11140-11144 (1995).
41. Horinouchi, S. & Weisblum, B. Nucleotide sequence and functional map of pC194, a plasmid that specifies inducible chloramphenicol resistance. J. Bacteriol. 150, 815-825 (1982).
42. Horton, R.M. In Vitro Recombination and Mutagenesis of DNA : SOEing Together Tailor-Made Genes. Methods Mol. Biol. 15, 251-261 (1993).
43. Podbielski, A., Spellerberg, B., Woischnik, M., Pohl, B. & Lutticken, R. Novel series of plasmid vectors for gene inactivation and expression analysis in group A streptococci (GAS). Gene 177, 137-147 (1996).
44. Husmann, L.K., Scott, J.R., Lindahl, G. & Stenberg, L. Expression of the Arp protein, a member of the M protein family, is not sufficient to inhibit phagocytosis of Streptococcus pyogenes. Infection and immunity 63, 345-348 (1995).
45. Gibson, D.G. et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods 6, 343-345 (2009).
46. Tangri S, et al. (“Rationally engineered therapeutic proteins with reduced immunogenicity” J Immunol. 2005 Mar 15;174(6):3187-96.
* * * *
Несмотря на то что предпочтительные варианты осуществления настоящего изобретения были показаны и описаны в данном документе, для специалиста в данной области будет очевидно, что такие варианты осуществления предоставлены только в качестве примера. Многочисленные вариации, изменения и замены теперь будут очевидны для специалиста в данной области без отступления от сути настоящего изобретения. Следует понимать, что различные альтернативные варианты вариантов осуществления настоящего изобретения, раскрытые в данном документе, можно применять при практическом осуществлении настоящего изобретения.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> THE BROAD INSTITUTE, INC.
MASSACHUSETTS INSTITUTE OF TECHNOLOGY
<120> ENGINEERING OF SYSTEMS, METHODS AND OPTIMIZED GUIDE COMPOSITIONS
FOR SEQUENCE MANIPULATION
<130> 44790.99.2047
<140> PCT/US2013/074819
<141> 2013-12-12
<150> 61/836,127
<151> 2013-06-17
<150> 61/835,931
<151> 2013-06-17
<150> 61/828,130
<151> 2013-05-28
<150> 61/819,803
<151> 2013-05-06
<150> 61/814,263
<151> 2013-04-20
<150> 61/806,375
<151> 2013-03-28
<150> 61/802,174
<151> 2013-03-15
<150> 61/791,409
<151> 2013-03-15
<150> 61/769,046
<151> 2013-02-25
<150> 61/758,468
<151> 2013-01-30
<150> 61/748,427
<151> 2013-01-02
<150> 61/736,527
<151> 2012-12-12
<160> 264
<170> PatentIn version 3.5
<210> 1
<211> 15
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 1
aggacgaagt cctaa 15
<210> 2
<211> 7
<212> PRT
<213> Simian virus 40
<400> 2
Pro Lys Lys Lys Arg Lys Val
1 5
<210> 3
<211> 16
<212> PRT
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Nucleoplasmin bipartite NLS sequence"
<400> 3
Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys
1 5 10 15
<210> 4
<211> 9
<212> PRT
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
C-myc NLS sequence"
<400> 4
Pro Ala Ala Lys Arg Val Lys Leu Asp
1 5
<210> 5
<211> 11
<212> PRT
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
C-myc NLS sequence"
<400> 5
Arg Gln Arg Arg Asn Glu Leu Lys Arg Ser Pro
1 5 10
<210> 6
<211> 38
<212> PRT
<213> Homo sapiens
<400> 6
Asn Gln Ser Ser Asn Phe Gly Pro Met Lys Gly Gly Asn Phe Gly Gly
1 5 10 15
Arg Ser Ser Gly Pro Tyr Gly Gly Gly Gly Gln Tyr Phe Ala Lys Pro
20 25 30
Arg Asn Gln Gly Gly Tyr
35
<210> 7
<211> 42
<212> PRT
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
IBB domain from importin-alpha sequence"
<400> 7
Arg Met Arg Ile Glx Phe Lys Asn Lys Gly Lys Asp Thr Ala Glu Leu
1 5 10 15
Arg Arg Arg Arg Val Glu Val Ser Val Glu Leu Arg Lys Ala Lys Lys
20 25 30
Asp Glu Gln Ile Leu Lys Arg Arg Asn Val
35 40
<210> 8
<211> 8
<212> PRT
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Myoma T protein sequence"
<400> 8
Val Ser Arg Lys Arg Pro Arg Pro
1 5
<210> 9
<211> 8
<212> PRT
<213> Unknown
<220>
<221> source
<223> /note="Description of Unknown:
Myoma T protein sequence"
<400> 9
Pro Pro Lys Lys Ala Arg Glu Asp
1 5
<210> 10
<211> 8
<212> PRT
<213> Homo sapiens
<400> 10
Pro Gln Pro Lys Lys Lys Pro Leu
1 5
<210> 11
<211> 12
<212> PRT
<213> Mus musculus
<400> 11
Ser Ala Leu Ile Lys Lys Lys Lys Lys Met Ala Pro
1 5 10
<210> 12
<211> 5
<212> PRT
<213> Influenza virus
<400> 12
Asp Arg Leu Arg Arg
1 5
<210> 13
<211> 7
<212> PRT
<213> Influenza virus
<400> 13
Pro Lys Gln Lys Lys Arg Lys
1 5
<210> 14
<211> 10
<212> PRT
<213> Hepatitus delta virus
<400> 14
Arg Lys Leu Lys Lys Lys Ile Lys Lys Leu
1 5 10
<210> 15
<211> 10
<212> PRT
<213> Mus musculus
<400> 15
Arg Glu Lys Lys Lys Phe Leu Lys Arg Arg
1 5 10
<210> 16
<211> 20
<212> PRT
<213> Homo sapiens
<400> 16
Lys Arg Lys Gly Asp Glu Val Asp Gly Val Asp Glu Val Ala Lys Lys
1 5 10 15
Lys Ser Lys Lys
20
<210> 17
<211> 17
<212> PRT
<213> Homo sapiens
<400> 17
Arg Lys Cys Leu Gln Ala Gly Met Asn Leu Glu Ala Arg Lys Thr Lys
1 5 10 15
Lys
<210> 18
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t or g
<220>
<221> modified_base
<222> (21)..(22)
<223> a, c, t, g, unknown or other
<400> 18
nnnnnnnnnn nnnnnnnnnn nnagaaw 27
<210> 19
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(12)
<223> a, c, t or g
<220>
<221> modified_base
<222> (13)..(14)
<223> a, c, t, g, unknown or other
<400> 19
nnnnnnnnnn nnnnagaaw 19
<210> 20
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t or g
<220>
<221> modified_base
<222> (21)..(22)
<223> a, c, t, g, unknown or other
<400> 20
nnnnnnnnnn nnnnnnnnnn nnagaaw 27
<210> 21
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(11)
<223> a, c, t or g
<220>
<221> modified_base
<222> (12)..(13)
<223> a, c, t, g, unknown or other
<400> 21
nnnnnnnnnn nnnagaaw 18
<210> 22
<211> 137
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 22
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatt tagaaataaa tcttgcagaa 60
gctacaaaga taaggcttca tgccgaaatc aacaccctgt cattttatgg cagggtgttt 120
tcgttattta atttttt 137
<210> 23
<211> 123
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 23
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcagaaat gcagaagcta caaagataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttcgt tatttaattt 120
ttt 123
<210> 24
<211> 110
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 24
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcagaaat gcagaagcta caaagataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttttt 110
<210> 25
<211> 102
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 25
nnnnnnnnnn nnnnnnnnnn gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcaac ttgaaaaagt ggcaccgagt cggtgctttt tt 102
<210> 26
<211> 88
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 26
nnnnnnnnnn nnnnnnnnnn gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcaac ttgaaaaagt gttttttt 88
<210> 27
<211> 76
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 27
nnnnnnnnnn nnnnnnnnnn gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcatt tttttt 76
<210> 28
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 28
guuuuagagc ua 12
<210> 29
<211> 33
<212> DNA
<213> Homo sapiens
<400> 29
ggacatcgat gtcacctcca atgactaggg tgg 33
<210> 30
<211> 33
<212> DNA
<213> Homo sapiens
<400> 30
cattggaggt gacatcgatg tcctccccat tgg 33
<210> 31
<211> 33
<212> DNA
<213> Homo sapiens
<400> 31
ggaagggcct gagtccgagc agaagaagaa ggg 33
<210> 32
<211> 33
<212> DNA
<213> Homo sapiens
<400> 32
ggtggcgaga ggggccgaga ttgggtgttc agg 33
<210> 33
<211> 33
<212> DNA
<213> Homo sapiens
<400> 33
atgcaggagg gtggcgagag gggccgagat tgg 33
<210> 34
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 34
aaaaccaccc ttctctctgg c 21
<210> 35
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 35
ggagattgga gacacggaga g 21
<210> 36
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 36
ctggaaagcc aatgcctgac 20
<210> 37
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
primer"
<400> 37
ggcagcaaac tccttgtcct 20
<210> 38
<211> 12
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 38
gttttagagc ta 12
<210> 39
<211> 335
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 39
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccg gaaccattca aaacagcata gcaagttaaa ataaggctag tccgttatca 300
acttgaaaaa gtggcaccga gtcggtgctt ttttt 335
<210> 40
<211> 423
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 40
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccg gtagtattaa gtattgtttt atggctgata aatttctttg aatttctcct 300
tgattatttg ttataaaagt tataaaataa tcttgttgga accattcaaa acagcatagc 360
aagttaaaat aaggctagtc cgttatcaac ttgaaaaagt ggcaccgagt cggtgctttt 420
ttt 423
<210> 41
<211> 339
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 41
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccg ggttttagag ctatgctgtt ttgaatggtc ccaaaacggg tcttcgagaa 300
gacgttttag agctatgctg ttttgaatgg tcccaaaac 339
<210> 42
<211> 309
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 42
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccg ggtcttcgag aagacctgtt ttagagctag aaatagcaag ttaaaataag 300
gctagtccg 309
<210> 43
<211> 1648
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 43
Met Asp Tyr Lys Asp His Asp Gly Asp Tyr Lys Asp His Asp Ile Asp
1 5 10 15
Tyr Lys Asp Asp Asp Asp Lys Met Ala Pro Lys Lys Lys Arg Lys Val
20 25 30
Gly Ile His Gly Val Pro Ala Ala Asp Lys Lys Tyr Ser Ile Gly Leu
35 40 45
Asp Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
50 55 60
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
65 70 75 80
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
85 90 95
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
100 105 110
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
115 120 125
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
130 135 140
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
145 150 155 160
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
165 170 175
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
180 185 190
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
195 200 205
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
210 215 220
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
225 230 235 240
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
245 250 255
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
260 265 270
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
275 280 285
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
290 295 300
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
305 310 315 320
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
325 330 335
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
340 345 350
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
355 360 365
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
370 375 380
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
385 390 395 400
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
405 410 415
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
420 425 430
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
435 440 445
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
450 455 460
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
465 470 475 480
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
485 490 495
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
500 505 510
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
515 520 525
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
530 535 540
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
545 550 555 560
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
565 570 575
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
580 585 590
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
595 600 605
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
610 615 620
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
625 630 635 640
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
645 650 655
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
660 665 670
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
675 680 685
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
690 695 700
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
705 710 715 720
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
725 730 735
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
740 745 750
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
755 760 765
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
770 775 780
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
785 790 795 800
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
805 810 815
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
820 825 830
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
835 840 845
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
850 855 860
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp His Ile
865 870 875 880
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
885 890 895
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser Glu
900 905 910
Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu Asn Ala
915 920 925
Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg
930 935 940
Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile Lys Arg Gln Leu
945 950 955 960
Val Glu Thr Arg Gln Ile Thr Lys His Val Ala Gln Ile Leu Asp Ser
965 970 975
Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp Lys Leu Ile Arg Glu Val
980 985 990
Lys Val Ile Thr Leu Lys Ser Lys Leu Val Ser Asp Phe Arg Lys Asp
995 1000 1005
Phe Gln Phe Tyr Lys Val Arg Glu Ile Asn Asn Tyr His His Ala
1010 1015 1020
His Asp Ala Tyr Leu Asn Ala Val Val Gly Thr Ala Leu Ile Lys
1025 1030 1035
Lys Tyr Pro Lys Leu Glu Ser Glu Phe Val Tyr Gly Asp Tyr Lys
1040 1045 1050
Val Tyr Asp Val Arg Lys Met Ile Ala Lys Ser Glu Gln Glu Ile
1055 1060 1065
Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser Asn Ile Met Asn
1070 1075 1080
Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu Ile Arg Lys
1085 1090 1095
Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile Val Trp
1100 1105 1110
Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser Met
1115 1120 1125
Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly
1130 1135 1140
Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu
1145 1150 1155
Ile Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe
1160 1165 1170
Asp Ser Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val
1175 1180 1185
Glu Lys Gly Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu
1190 1195 1200
Gly Ile Thr Ile Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile
1205 1210 1215
Asp Phe Leu Glu Ala Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu
1220 1225 1230
Ile Ile Lys Leu Pro Lys Tyr Ser Leu Phe Glu Leu Glu Asn Gly
1235 1240 1245
Arg Lys Arg Met Leu Ala Ser Ala Gly Glu Leu Gln Lys Gly Asn
1250 1255 1260
Glu Leu Ala Leu Pro Ser Lys Tyr Val Asn Phe Leu Tyr Leu Ala
1265 1270 1275
Ser His Tyr Glu Lys Leu Lys Gly Ser Pro Glu Asp Asn Glu Gln
1280 1285 1290
Lys Gln Leu Phe Val Glu Gln His Lys His Tyr Leu Asp Glu Ile
1295 1300 1305
Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val Ile Leu Ala Asp
1310 1315 1320
Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys His Arg Asp
1325 1330 1335
Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu Phe Thr
1340 1345 1350
Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp Thr
1355 1360 1365
Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp
1370 1375 1380
Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg
1385 1390 1395
Ile Asp Leu Ser Gln Leu Gly Gly Asp Ala Ala Ala Val Ser Lys
1400 1405 1410
Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu Val Glu Leu
1415 1420 1425
Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly Glu Gly
1430 1435 1440
Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile Cys
1445 1450 1455
Thr Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr
1460 1465 1470
Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg Tyr Pro Asp His Met
1475 1480 1485
Lys Gln His Asp Phe Phe Lys Ser Ala Met Pro Glu Gly Tyr Val
1490 1495 1500
Gln Glu Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys Thr
1505 1510 1515
Arg Ala Glu Val Lys Phe Glu Gly Asp Thr Leu Val Asn Arg Ile
1520 1525 1530
Glu Leu Lys Gly Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly
1535 1540 1545
His Lys Leu Glu Tyr Asn Tyr Asn Ser His Asn Val Tyr Ile Met
1550 1555 1560
Ala Asp Lys Gln Lys Asn Gly Ile Lys Val Asn Phe Lys Ile Arg
1565 1570 1575
His Asn Ile Glu Asp Gly Ser Val Gln Leu Ala Asp His Tyr Gln
1580 1585 1590
Gln Asn Thr Pro Ile Gly Asp Gly Pro Val Leu Leu Pro Asp Asn
1595 1600 1605
His Tyr Leu Ser Thr Gln Ser Ala Leu Ser Lys Asp Pro Asn Glu
1610 1615 1620
Lys Arg Asp His Met Val Leu Leu Glu Phe Val Thr Ala Ala Gly
1625 1630 1635
Ile Thr Leu Gly Met Asp Glu Leu Tyr Lys
1640 1645
<210> 44
<211> 1625
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 44
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
Ala Ala Ala Val Ser Lys Gly Glu Glu Leu Phe Thr Gly Val Val
1370 1375 1380
Pro Ile Leu Val Glu Leu Asp Gly Asp Val Asn Gly His Lys Phe
1385 1390 1395
Ser Val Ser Gly Glu Gly Glu Gly Asp Ala Thr Tyr Gly Lys Leu
1400 1405 1410
Thr Leu Lys Phe Ile Cys Thr Thr Gly Lys Leu Pro Val Pro Trp
1415 1420 1425
Pro Thr Leu Val Thr Thr Leu Thr Tyr Gly Val Gln Cys Phe Ser
1430 1435 1440
Arg Tyr Pro Asp His Met Lys Gln His Asp Phe Phe Lys Ser Ala
1445 1450 1455
Met Pro Glu Gly Tyr Val Gln Glu Arg Thr Ile Phe Phe Lys Asp
1460 1465 1470
Asp Gly Asn Tyr Lys Thr Arg Ala Glu Val Lys Phe Glu Gly Asp
1475 1480 1485
Thr Leu Val Asn Arg Ile Glu Leu Lys Gly Ile Asp Phe Lys Glu
1490 1495 1500
Asp Gly Asn Ile Leu Gly His Lys Leu Glu Tyr Asn Tyr Asn Ser
1505 1510 1515
His Asn Val Tyr Ile Met Ala Asp Lys Gln Lys Asn Gly Ile Lys
1520 1525 1530
Val Asn Phe Lys Ile Arg His Asn Ile Glu Asp Gly Ser Val Gln
1535 1540 1545
Leu Ala Asp His Tyr Gln Gln Asn Thr Pro Ile Gly Asp Gly Pro
1550 1555 1560
Val Leu Leu Pro Asp Asn His Tyr Leu Ser Thr Gln Ser Ala Leu
1565 1570 1575
Ser Lys Asp Pro Asn Glu Lys Arg Asp His Met Val Leu Leu Glu
1580 1585 1590
Phe Val Thr Ala Ala Gly Ile Thr Leu Gly Met Asp Glu Leu Tyr
1595 1600 1605
Lys Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys
1610 1615 1620
Lys Lys
1625
<210> 45
<211> 1664
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 45
Met Asp Tyr Lys Asp His Asp Gly Asp Tyr Lys Asp His Asp Ile Asp
1 5 10 15
Tyr Lys Asp Asp Asp Asp Lys Met Ala Pro Lys Lys Lys Arg Lys Val
20 25 30
Gly Ile His Gly Val Pro Ala Ala Asp Lys Lys Tyr Ser Ile Gly Leu
35 40 45
Asp Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
50 55 60
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
65 70 75 80
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
85 90 95
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
100 105 110
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
115 120 125
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
130 135 140
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
145 150 155 160
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
165 170 175
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
180 185 190
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
195 200 205
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
210 215 220
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
225 230 235 240
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
245 250 255
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
260 265 270
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
275 280 285
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
290 295 300
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
305 310 315 320
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
325 330 335
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
340 345 350
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
355 360 365
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
370 375 380
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
385 390 395 400
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
405 410 415
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
420 425 430
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
435 440 445
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
450 455 460
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
465 470 475 480
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
485 490 495
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
500 505 510
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
515 520 525
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
530 535 540
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
545 550 555 560
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
565 570 575
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
580 585 590
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
595 600 605
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
610 615 620
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
625 630 635 640
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
645 650 655
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
660 665 670
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
675 680 685
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
690 695 700
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
705 710 715 720
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
725 730 735
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
740 745 750
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
755 760 765
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
770 775 780
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
785 790 795 800
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
805 810 815
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
820 825 830
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
835 840 845
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
850 855 860
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp His Ile
865 870 875 880
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
885 890 895
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser Glu
900 905 910
Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu Asn Ala
915 920 925
Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg
930 935 940
Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile Lys Arg Gln Leu
945 950 955 960
Val Glu Thr Arg Gln Ile Thr Lys His Val Ala Gln Ile Leu Asp Ser
965 970 975
Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp Lys Leu Ile Arg Glu Val
980 985 990
Lys Val Ile Thr Leu Lys Ser Lys Leu Val Ser Asp Phe Arg Lys Asp
995 1000 1005
Phe Gln Phe Tyr Lys Val Arg Glu Ile Asn Asn Tyr His His Ala
1010 1015 1020
His Asp Ala Tyr Leu Asn Ala Val Val Gly Thr Ala Leu Ile Lys
1025 1030 1035
Lys Tyr Pro Lys Leu Glu Ser Glu Phe Val Tyr Gly Asp Tyr Lys
1040 1045 1050
Val Tyr Asp Val Arg Lys Met Ile Ala Lys Ser Glu Gln Glu Ile
1055 1060 1065
Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser Asn Ile Met Asn
1070 1075 1080
Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu Ile Arg Lys
1085 1090 1095
Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile Val Trp
1100 1105 1110
Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser Met
1115 1120 1125
Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly
1130 1135 1140
Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu
1145 1150 1155
Ile Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe
1160 1165 1170
Asp Ser Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val
1175 1180 1185
Glu Lys Gly Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu
1190 1195 1200
Gly Ile Thr Ile Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile
1205 1210 1215
Asp Phe Leu Glu Ala Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu
1220 1225 1230
Ile Ile Lys Leu Pro Lys Tyr Ser Leu Phe Glu Leu Glu Asn Gly
1235 1240 1245
Arg Lys Arg Met Leu Ala Ser Ala Gly Glu Leu Gln Lys Gly Asn
1250 1255 1260
Glu Leu Ala Leu Pro Ser Lys Tyr Val Asn Phe Leu Tyr Leu Ala
1265 1270 1275
Ser His Tyr Glu Lys Leu Lys Gly Ser Pro Glu Asp Asn Glu Gln
1280 1285 1290
Lys Gln Leu Phe Val Glu Gln His Lys His Tyr Leu Asp Glu Ile
1295 1300 1305
Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val Ile Leu Ala Asp
1310 1315 1320
Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys His Arg Asp
1325 1330 1335
Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu Phe Thr
1340 1345 1350
Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp Thr
1355 1360 1365
Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp
1370 1375 1380
Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg
1385 1390 1395
Ile Asp Leu Ser Gln Leu Gly Gly Asp Ala Ala Ala Val Ser Lys
1400 1405 1410
Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu Val Glu Leu
1415 1420 1425
Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly Glu Gly
1430 1435 1440
Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile Cys
1445 1450 1455
Thr Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr
1460 1465 1470
Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg Tyr Pro Asp His Met
1475 1480 1485
Lys Gln His Asp Phe Phe Lys Ser Ala Met Pro Glu Gly Tyr Val
1490 1495 1500
Gln Glu Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys Thr
1505 1510 1515
Arg Ala Glu Val Lys Phe Glu Gly Asp Thr Leu Val Asn Arg Ile
1520 1525 1530
Glu Leu Lys Gly Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly
1535 1540 1545
His Lys Leu Glu Tyr Asn Tyr Asn Ser His Asn Val Tyr Ile Met
1550 1555 1560
Ala Asp Lys Gln Lys Asn Gly Ile Lys Val Asn Phe Lys Ile Arg
1565 1570 1575
His Asn Ile Glu Asp Gly Ser Val Gln Leu Ala Asp His Tyr Gln
1580 1585 1590
Gln Asn Thr Pro Ile Gly Asp Gly Pro Val Leu Leu Pro Asp Asn
1595 1600 1605
His Tyr Leu Ser Thr Gln Ser Ala Leu Ser Lys Asp Pro Asn Glu
1610 1615 1620
Lys Arg Asp His Met Val Leu Leu Glu Phe Val Thr Ala Ala Gly
1625 1630 1635
Ile Thr Leu Gly Met Asp Glu Leu Tyr Lys Lys Arg Pro Ala Ala
1640 1645 1650
Thr Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys
1655 1660
<210> 46
<211> 1423
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 46
Met Asp Tyr Lys Asp His Asp Gly Asp Tyr Lys Asp His Asp Ile Asp
1 5 10 15
Tyr Lys Asp Asp Asp Asp Lys Met Ala Pro Lys Lys Lys Arg Lys Val
20 25 30
Gly Ile His Gly Val Pro Ala Ala Asp Lys Lys Tyr Ser Ile Gly Leu
35 40 45
Asp Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
50 55 60
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
65 70 75 80
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
85 90 95
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
100 105 110
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
115 120 125
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
130 135 140
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
145 150 155 160
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
165 170 175
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
180 185 190
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
195 200 205
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
210 215 220
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
225 230 235 240
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
245 250 255
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
260 265 270
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
275 280 285
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
290 295 300
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
305 310 315 320
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
325 330 335
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
340 345 350
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
355 360 365
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
370 375 380
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
385 390 395 400
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
405 410 415
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
420 425 430
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
435 440 445
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
450 455 460
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
465 470 475 480
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
485 490 495
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
500 505 510
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
515 520 525
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
530 535 540
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
545 550 555 560
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
565 570 575
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
580 585 590
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
595 600 605
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
610 615 620
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
625 630 635 640
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
645 650 655
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
660 665 670
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
675 680 685
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
690 695 700
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
705 710 715 720
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
725 730 735
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
740 745 750
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
755 760 765
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
770 775 780
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
785 790 795 800
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
805 810 815
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
820 825 830
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
835 840 845
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
850 855 860
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp His Ile
865 870 875 880
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
885 890 895
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser Glu
900 905 910
Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu Asn Ala
915 920 925
Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg
930 935 940
Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile Lys Arg Gln Leu
945 950 955 960
Val Glu Thr Arg Gln Ile Thr Lys His Val Ala Gln Ile Leu Asp Ser
965 970 975
Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp Lys Leu Ile Arg Glu Val
980 985 990
Lys Val Ile Thr Leu Lys Ser Lys Leu Val Ser Asp Phe Arg Lys Asp
995 1000 1005
Phe Gln Phe Tyr Lys Val Arg Glu Ile Asn Asn Tyr His His Ala
1010 1015 1020
His Asp Ala Tyr Leu Asn Ala Val Val Gly Thr Ala Leu Ile Lys
1025 1030 1035
Lys Tyr Pro Lys Leu Glu Ser Glu Phe Val Tyr Gly Asp Tyr Lys
1040 1045 1050
Val Tyr Asp Val Arg Lys Met Ile Ala Lys Ser Glu Gln Glu Ile
1055 1060 1065
Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser Asn Ile Met Asn
1070 1075 1080
Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu Ile Arg Lys
1085 1090 1095
Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile Val Trp
1100 1105 1110
Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser Met
1115 1120 1125
Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly
1130 1135 1140
Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu
1145 1150 1155
Ile Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe
1160 1165 1170
Asp Ser Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val
1175 1180 1185
Glu Lys Gly Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu
1190 1195 1200
Gly Ile Thr Ile Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile
1205 1210 1215
Asp Phe Leu Glu Ala Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu
1220 1225 1230
Ile Ile Lys Leu Pro Lys Tyr Ser Leu Phe Glu Leu Glu Asn Gly
1235 1240 1245
Arg Lys Arg Met Leu Ala Ser Ala Gly Glu Leu Gln Lys Gly Asn
1250 1255 1260
Glu Leu Ala Leu Pro Ser Lys Tyr Val Asn Phe Leu Tyr Leu Ala
1265 1270 1275
Ser His Tyr Glu Lys Leu Lys Gly Ser Pro Glu Asp Asn Glu Gln
1280 1285 1290
Lys Gln Leu Phe Val Glu Gln His Lys His Tyr Leu Asp Glu Ile
1295 1300 1305
Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val Ile Leu Ala Asp
1310 1315 1320
Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys His Arg Asp
1325 1330 1335
Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu Phe Thr
1340 1345 1350
Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp Thr
1355 1360 1365
Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp
1370 1375 1380
Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg
1385 1390 1395
Ile Asp Leu Ser Gln Leu Gly Gly Asp Lys Arg Pro Ala Ala Thr
1400 1405 1410
Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys
1415 1420
<210> 47
<211> 483
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 47
Met Phe Leu Phe Leu Ser Leu Thr Ser Phe Leu Ser Ser Ser Arg Thr
1 5 10 15
Leu Val Ser Lys Gly Glu Glu Asp Asn Met Ala Ile Ile Lys Glu Phe
20 25 30
Met Arg Phe Lys Val His Met Glu Gly Ser Val Asn Gly His Glu Phe
35 40 45
Glu Ile Glu Gly Glu Gly Glu Gly Arg Pro Tyr Glu Gly Thr Gln Thr
50 55 60
Ala Lys Leu Lys Val Thr Lys Gly Gly Pro Leu Pro Phe Ala Trp Asp
65 70 75 80
Ile Leu Ser Pro Gln Phe Met Tyr Gly Ser Lys Ala Tyr Val Lys His
85 90 95
Pro Ala Asp Ile Pro Asp Tyr Leu Lys Leu Ser Phe Pro Glu Gly Phe
100 105 110
Lys Trp Glu Arg Val Met Asn Phe Glu Asp Gly Gly Val Val Thr Val
115 120 125
Thr Gln Asp Ser Ser Leu Gln Asp Gly Glu Phe Ile Tyr Lys Val Lys
130 135 140
Leu Arg Gly Thr Asn Phe Pro Ser Asp Gly Pro Val Met Gln Lys Lys
145 150 155 160
Thr Met Gly Trp Glu Ala Ser Ser Glu Arg Met Tyr Pro Glu Asp Gly
165 170 175
Ala Leu Lys Gly Glu Ile Lys Gln Arg Leu Lys Leu Lys Asp Gly Gly
180 185 190
His Tyr Asp Ala Glu Val Lys Thr Thr Tyr Lys Ala Lys Lys Pro Val
195 200 205
Gln Leu Pro Gly Ala Tyr Asn Val Asn Ile Lys Leu Asp Ile Thr Ser
210 215 220
His Asn Glu Asp Tyr Thr Ile Val Glu Gln Tyr Glu Arg Ala Glu Gly
225 230 235 240
Arg His Ser Thr Gly Gly Met Asp Glu Leu Tyr Lys Gly Ser Lys Gln
245 250 255
Leu Glu Glu Leu Leu Ser Thr Ser Phe Asp Ile Gln Phe Asn Asp Leu
260 265 270
Thr Leu Leu Glu Thr Ala Phe Thr His Thr Ser Tyr Ala Asn Glu His
275 280 285
Arg Leu Leu Asn Val Ser His Asn Glu Arg Leu Glu Phe Leu Gly Asp
290 295 300
Ala Val Leu Gln Leu Ile Ile Ser Glu Tyr Leu Phe Ala Lys Tyr Pro
305 310 315 320
Lys Lys Thr Glu Gly Asp Met Ser Lys Leu Arg Ser Met Ile Val Arg
325 330 335
Glu Glu Ser Leu Ala Gly Phe Ser Arg Phe Cys Ser Phe Asp Ala Tyr
340 345 350
Ile Lys Leu Gly Lys Gly Glu Glu Lys Ser Gly Gly Arg Arg Arg Asp
355 360 365
Thr Ile Leu Gly Asp Leu Phe Glu Ala Phe Leu Gly Ala Leu Leu Leu
370 375 380
Asp Lys Gly Ile Asp Ala Val Arg Arg Phe Leu Lys Gln Val Met Ile
385 390 395 400
Pro Gln Val Glu Lys Gly Asn Phe Glu Arg Val Lys Asp Tyr Lys Thr
405 410 415
Cys Leu Gln Glu Phe Leu Gln Thr Lys Gly Asp Val Ala Ile Asp Tyr
420 425 430
Gln Val Ile Ser Glu Lys Gly Pro Ala His Ala Lys Gln Phe Glu Val
435 440 445
Ser Ile Val Val Asn Gly Ala Val Leu Ser Lys Gly Leu Gly Lys Ser
450 455 460
Lys Lys Leu Ala Glu Gln Asp Ala Ala Lys Asn Ala Leu Ala Gln Leu
465 470 475 480
Ser Glu Val
<210> 48
<211> 483
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 48
Met Lys Gln Leu Glu Glu Leu Leu Ser Thr Ser Phe Asp Ile Gln Phe
1 5 10 15
Asn Asp Leu Thr Leu Leu Glu Thr Ala Phe Thr His Thr Ser Tyr Ala
20 25 30
Asn Glu His Arg Leu Leu Asn Val Ser His Asn Glu Arg Leu Glu Phe
35 40 45
Leu Gly Asp Ala Val Leu Gln Leu Ile Ile Ser Glu Tyr Leu Phe Ala
50 55 60
Lys Tyr Pro Lys Lys Thr Glu Gly Asp Met Ser Lys Leu Arg Ser Met
65 70 75 80
Ile Val Arg Glu Glu Ser Leu Ala Gly Phe Ser Arg Phe Cys Ser Phe
85 90 95
Asp Ala Tyr Ile Lys Leu Gly Lys Gly Glu Glu Lys Ser Gly Gly Arg
100 105 110
Arg Arg Asp Thr Ile Leu Gly Asp Leu Phe Glu Ala Phe Leu Gly Ala
115 120 125
Leu Leu Leu Asp Lys Gly Ile Asp Ala Val Arg Arg Phe Leu Lys Gln
130 135 140
Val Met Ile Pro Gln Val Glu Lys Gly Asn Phe Glu Arg Val Lys Asp
145 150 155 160
Tyr Lys Thr Cys Leu Gln Glu Phe Leu Gln Thr Lys Gly Asp Val Ala
165 170 175
Ile Asp Tyr Gln Val Ile Ser Glu Lys Gly Pro Ala His Ala Lys Gln
180 185 190
Phe Glu Val Ser Ile Val Val Asn Gly Ala Val Leu Ser Lys Gly Leu
195 200 205
Gly Lys Ser Lys Lys Leu Ala Glu Gln Asp Ala Ala Lys Asn Ala Leu
210 215 220
Ala Gln Leu Ser Glu Val Gly Ser Val Ser Lys Gly Glu Glu Asp Asn
225 230 235 240
Met Ala Ile Ile Lys Glu Phe Met Arg Phe Lys Val His Met Glu Gly
245 250 255
Ser Val Asn Gly His Glu Phe Glu Ile Glu Gly Glu Gly Glu Gly Arg
260 265 270
Pro Tyr Glu Gly Thr Gln Thr Ala Lys Leu Lys Val Thr Lys Gly Gly
275 280 285
Pro Leu Pro Phe Ala Trp Asp Ile Leu Ser Pro Gln Phe Met Tyr Gly
290 295 300
Ser Lys Ala Tyr Val Lys His Pro Ala Asp Ile Pro Asp Tyr Leu Lys
305 310 315 320
Leu Ser Phe Pro Glu Gly Phe Lys Trp Glu Arg Val Met Asn Phe Glu
325 330 335
Asp Gly Gly Val Val Thr Val Thr Gln Asp Ser Ser Leu Gln Asp Gly
340 345 350
Glu Phe Ile Tyr Lys Val Lys Leu Arg Gly Thr Asn Phe Pro Ser Asp
355 360 365
Gly Pro Val Met Gln Lys Lys Thr Met Gly Trp Glu Ala Ser Ser Glu
370 375 380
Arg Met Tyr Pro Glu Asp Gly Ala Leu Lys Gly Glu Ile Lys Gln Arg
385 390 395 400
Leu Lys Leu Lys Asp Gly Gly His Tyr Asp Ala Glu Val Lys Thr Thr
405 410 415
Tyr Lys Ala Lys Lys Pro Val Gln Leu Pro Gly Ala Tyr Asn Val Asn
420 425 430
Ile Lys Leu Asp Ile Thr Ser His Asn Glu Asp Tyr Thr Ile Val Glu
435 440 445
Gln Tyr Glu Arg Ala Glu Gly Arg His Ser Thr Gly Gly Met Asp Glu
450 455 460
Leu Tyr Lys Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys
465 470 475 480
Lys Lys Lys
<210> 49
<211> 1423
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 49
Met Asp Tyr Lys Asp His Asp Gly Asp Tyr Lys Asp His Asp Ile Asp
1 5 10 15
Tyr Lys Asp Asp Asp Asp Lys Met Ala Pro Lys Lys Lys Arg Lys Val
20 25 30
Gly Ile His Gly Val Pro Ala Ala Asp Lys Lys Tyr Ser Ile Gly Leu
35 40 45
Ala Ile Gly Thr Asn Ser Val Gly Trp Ala Val Ile Thr Asp Glu Tyr
50 55 60
Lys Val Pro Ser Lys Lys Phe Lys Val Leu Gly Asn Thr Asp Arg His
65 70 75 80
Ser Ile Lys Lys Asn Leu Ile Gly Ala Leu Leu Phe Asp Ser Gly Glu
85 90 95
Thr Ala Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr
100 105 110
Arg Arg Lys Asn Arg Ile Cys Tyr Leu Gln Glu Ile Phe Ser Asn Glu
115 120 125
Met Ala Lys Val Asp Asp Ser Phe Phe His Arg Leu Glu Glu Ser Phe
130 135 140
Leu Val Glu Glu Asp Lys Lys His Glu Arg His Pro Ile Phe Gly Asn
145 150 155 160
Ile Val Asp Glu Val Ala Tyr His Glu Lys Tyr Pro Thr Ile Tyr His
165 170 175
Leu Arg Lys Lys Leu Val Asp Ser Thr Asp Lys Ala Asp Leu Arg Leu
180 185 190
Ile Tyr Leu Ala Leu Ala His Met Ile Lys Phe Arg Gly His Phe Leu
195 200 205
Ile Glu Gly Asp Leu Asn Pro Asp Asn Ser Asp Val Asp Lys Leu Phe
210 215 220
Ile Gln Leu Val Gln Thr Tyr Asn Gln Leu Phe Glu Glu Asn Pro Ile
225 230 235 240
Asn Ala Ser Gly Val Asp Ala Lys Ala Ile Leu Ser Ala Arg Leu Ser
245 250 255
Lys Ser Arg Arg Leu Glu Asn Leu Ile Ala Gln Leu Pro Gly Glu Lys
260 265 270
Lys Asn Gly Leu Phe Gly Asn Leu Ile Ala Leu Ser Leu Gly Leu Thr
275 280 285
Pro Asn Phe Lys Ser Asn Phe Asp Leu Ala Glu Asp Ala Lys Leu Gln
290 295 300
Leu Ser Lys Asp Thr Tyr Asp Asp Asp Leu Asp Asn Leu Leu Ala Gln
305 310 315 320
Ile Gly Asp Gln Tyr Ala Asp Leu Phe Leu Ala Ala Lys Asn Leu Ser
325 330 335
Asp Ala Ile Leu Leu Ser Asp Ile Leu Arg Val Asn Thr Glu Ile Thr
340 345 350
Lys Ala Pro Leu Ser Ala Ser Met Ile Lys Arg Tyr Asp Glu His His
355 360 365
Gln Asp Leu Thr Leu Leu Lys Ala Leu Val Arg Gln Gln Leu Pro Glu
370 375 380
Lys Tyr Lys Glu Ile Phe Phe Asp Gln Ser Lys Asn Gly Tyr Ala Gly
385 390 395 400
Tyr Ile Asp Gly Gly Ala Ser Gln Glu Glu Phe Tyr Lys Phe Ile Lys
405 410 415
Pro Ile Leu Glu Lys Met Asp Gly Thr Glu Glu Leu Leu Val Lys Leu
420 425 430
Asn Arg Glu Asp Leu Leu Arg Lys Gln Arg Thr Phe Asp Asn Gly Ser
435 440 445
Ile Pro His Gln Ile His Leu Gly Glu Leu His Ala Ile Leu Arg Arg
450 455 460
Gln Glu Asp Phe Tyr Pro Phe Leu Lys Asp Asn Arg Glu Lys Ile Glu
465 470 475 480
Lys Ile Leu Thr Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Ala Arg
485 490 495
Gly Asn Ser Arg Phe Ala Trp Met Thr Arg Lys Ser Glu Glu Thr Ile
500 505 510
Thr Pro Trp Asn Phe Glu Glu Val Val Asp Lys Gly Ala Ser Ala Gln
515 520 525
Ser Phe Ile Glu Arg Met Thr Asn Phe Asp Lys Asn Leu Pro Asn Glu
530 535 540
Lys Val Leu Pro Lys His Ser Leu Leu Tyr Glu Tyr Phe Thr Val Tyr
545 550 555 560
Asn Glu Leu Thr Lys Val Lys Tyr Val Thr Glu Gly Met Arg Lys Pro
565 570 575
Ala Phe Leu Ser Gly Glu Gln Lys Lys Ala Ile Val Asp Leu Leu Phe
580 585 590
Lys Thr Asn Arg Lys Val Thr Val Lys Gln Leu Lys Glu Asp Tyr Phe
595 600 605
Lys Lys Ile Glu Cys Phe Asp Ser Val Glu Ile Ser Gly Val Glu Asp
610 615 620
Arg Phe Asn Ala Ser Leu Gly Thr Tyr His Asp Leu Leu Lys Ile Ile
625 630 635 640
Lys Asp Lys Asp Phe Leu Asp Asn Glu Glu Asn Glu Asp Ile Leu Glu
645 650 655
Asp Ile Val Leu Thr Leu Thr Leu Phe Glu Asp Arg Glu Met Ile Glu
660 665 670
Glu Arg Leu Lys Thr Tyr Ala His Leu Phe Asp Asp Lys Val Met Lys
675 680 685
Gln Leu Lys Arg Arg Arg Tyr Thr Gly Trp Gly Arg Leu Ser Arg Lys
690 695 700
Leu Ile Asn Gly Ile Arg Asp Lys Gln Ser Gly Lys Thr Ile Leu Asp
705 710 715 720
Phe Leu Lys Ser Asp Gly Phe Ala Asn Arg Asn Phe Met Gln Leu Ile
725 730 735
His Asp Asp Ser Leu Thr Phe Lys Glu Asp Ile Gln Lys Ala Gln Val
740 745 750
Ser Gly Gln Gly Asp Ser Leu His Glu His Ile Ala Asn Leu Ala Gly
755 760 765
Ser Pro Ala Ile Lys Lys Gly Ile Leu Gln Thr Val Lys Val Val Asp
770 775 780
Glu Leu Val Lys Val Met Gly Arg His Lys Pro Glu Asn Ile Val Ile
785 790 795 800
Glu Met Ala Arg Glu Asn Gln Thr Thr Gln Lys Gly Gln Lys Asn Ser
805 810 815
Arg Glu Arg Met Lys Arg Ile Glu Glu Gly Ile Lys Glu Leu Gly Ser
820 825 830
Gln Ile Leu Lys Glu His Pro Val Glu Asn Thr Gln Leu Gln Asn Glu
835 840 845
Lys Leu Tyr Leu Tyr Tyr Leu Gln Asn Gly Arg Asp Met Tyr Val Asp
850 855 860
Gln Glu Leu Asp Ile Asn Arg Leu Ser Asp Tyr Asp Val Asp His Ile
865 870 875 880
Val Pro Gln Ser Phe Leu Lys Asp Asp Ser Ile Asp Asn Lys Val Leu
885 890 895
Thr Arg Ser Asp Lys Asn Arg Gly Lys Ser Asp Asn Val Pro Ser Glu
900 905 910
Glu Val Val Lys Lys Met Lys Asn Tyr Trp Arg Gln Leu Leu Asn Ala
915 920 925
Lys Leu Ile Thr Gln Arg Lys Phe Asp Asn Leu Thr Lys Ala Glu Arg
930 935 940
Gly Gly Leu Ser Glu Leu Asp Lys Ala Gly Phe Ile Lys Arg Gln Leu
945 950 955 960
Val Glu Thr Arg Gln Ile Thr Lys His Val Ala Gln Ile Leu Asp Ser
965 970 975
Arg Met Asn Thr Lys Tyr Asp Glu Asn Asp Lys Leu Ile Arg Glu Val
980 985 990
Lys Val Ile Thr Leu Lys Ser Lys Leu Val Ser Asp Phe Arg Lys Asp
995 1000 1005
Phe Gln Phe Tyr Lys Val Arg Glu Ile Asn Asn Tyr His His Ala
1010 1015 1020
His Asp Ala Tyr Leu Asn Ala Val Val Gly Thr Ala Leu Ile Lys
1025 1030 1035
Lys Tyr Pro Lys Leu Glu Ser Glu Phe Val Tyr Gly Asp Tyr Lys
1040 1045 1050
Val Tyr Asp Val Arg Lys Met Ile Ala Lys Ser Glu Gln Glu Ile
1055 1060 1065
Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser Asn Ile Met Asn
1070 1075 1080
Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu Ile Arg Lys
1085 1090 1095
Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile Val Trp
1100 1105 1110
Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser Met
1115 1120 1125
Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly
1130 1135 1140
Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu
1145 1150 1155
Ile Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe
1160 1165 1170
Asp Ser Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val
1175 1180 1185
Glu Lys Gly Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu
1190 1195 1200
Gly Ile Thr Ile Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile
1205 1210 1215
Asp Phe Leu Glu Ala Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu
1220 1225 1230
Ile Ile Lys Leu Pro Lys Tyr Ser Leu Phe Glu Leu Glu Asn Gly
1235 1240 1245
Arg Lys Arg Met Leu Ala Ser Ala Gly Glu Leu Gln Lys Gly Asn
1250 1255 1260
Glu Leu Ala Leu Pro Ser Lys Tyr Val Asn Phe Leu Tyr Leu Ala
1265 1270 1275
Ser His Tyr Glu Lys Leu Lys Gly Ser Pro Glu Asp Asn Glu Gln
1280 1285 1290
Lys Gln Leu Phe Val Glu Gln His Lys His Tyr Leu Asp Glu Ile
1295 1300 1305
Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val Ile Leu Ala Asp
1310 1315 1320
Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys His Arg Asp
1325 1330 1335
Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu Phe Thr
1340 1345 1350
Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp Thr
1355 1360 1365
Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp
1370 1375 1380
Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg
1385 1390 1395
Ile Asp Leu Ser Gln Leu Gly Gly Asp Lys Arg Pro Ala Ala Thr
1400 1405 1410
Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys
1415 1420
<210> 50
<211> 2012
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 50
gaatgctgcc ctcagacccg cttcctccct gtccttgtct gtccaaggag aatgaggtct 60
cactggtgga tttcggacta ccctgaggag ctggcacctg agggacaagg ccccccacct 120
gcccagctcc agcctctgat gaggggtggg agagagctac atgaggttgc taagaaagcc 180
tcccctgaag gagaccacac agtgtgtgag gttggagtct ctagcagcgg gttctgtgcc 240
cccagggata gtctggctgt ccaggcactg ctcttgatat aaacaccacc tcctagttat 300
gaaaccatgc ccattctgcc tctctgtatg gaaaagagca tggggctggc ccgtggggtg 360
gtgtccactt taggccctgt gggagatcat gggaacccac gcagtgggtc ataggctctc 420
tcatttacta ctcacatcca ctctgtgaag aagcgattat gatctctcct ctagaaactc 480
gtagagtccc atgtctgccg gcttccagag cctgcactcc tccaccttgg cttggctttg 540
ctggggctag aggagctagg atgcacagca gctctgtgac cctttgtttg agaggaacag 600
gaaaaccacc cttctctctg gcccactgtg tcctcttcct gccctgccat ccccttctgt 660
gaatgttaga cccatgggag cagctggtca gaggggaccc cggcctgggg cccctaaccc 720
tatgtagcct cagtcttccc atcaggctct cagctcagcc tgagtgttga ggccccagtg 780
gctgctctgg gggcctcctg agtttctcat ctgtgcccct ccctccctgg cccaggtgaa 840
ggtgtggttc cagaaccgga ggacaaagta caaacggcag aagctggagg aggaagggcc 900
tgagtccgag cagaagaaga agggctccca tcacatcaac cggtggcgca ttgccacgaa 960
gcaggccaat ggggaggaca tcgatgtcac ctccaatgac aagcttgcta gcggtgggca 1020
accacaaacc cacgagggca gagtgctgct tgctgctggc caggcccctg cgtgggccca 1080
agctggactc tggccactcc ctggccaggc tttggggagg cctggagtca tggccccaca 1140
gggcttgaag cccggggccg ccattgacag agggacaagc aatgggctgg ctgaggcctg 1200
ggaccacttg gccttctcct cggagagcct gcctgcctgg gcgggcccgc ccgccaccgc 1260
agcctcccag ctgctctccg tgtctccaat ctcccttttg ttttgatgca tttctgtttt 1320
aatttatttt ccaggcacca ctgtagttta gtgatcccca gtgtccccct tccctatggg 1380
aataataaaa gtctctctct taatgacacg ggcatccagc tccagcccca gagcctgggg 1440
tggtagattc cggctctgag ggccagtggg ggctggtaga gcaaacgcgt tcagggcctg 1500
ggagcctggg gtggggtact ggtggagggg gtcaagggta attcattaac tcctctcttt 1560
tgttggggga ccctggtctc tacctccagc tccacagcag gagaaacagg ctagacatag 1620
ggaagggcca tcctgtatct tgagggagga caggcccagg tctttcttaa cgtattgaga 1680
ggtgggaatc aggcccaggt agttcaatgg gagagggaga gtgcttccct ctgcctagag 1740
actctggtgg cttctccagt tgaggagaaa ccagaggaaa ggggaggatt ggggtctggg 1800
ggagggaaca ccattcacaa aggctgacgg ttccagtccg aagtcgtggg cccaccagga 1860
tgctcacctg tccttggaga accgctgggc aggttgagac tgcagagaca gggcttaagg 1920
ctgagcctgc aaccagtccc cagtgactca gggcctcctc agcccaagaa agagcaacgt 1980
gccagggccc gctgagctct tgtgttcacc tg 2012
<210> 51
<211> 1153
<212> PRT
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polypeptide"
<400> 51
Met Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys Lys
1 5 10 15
Lys Ser Asp Leu Val Leu Gly Leu Asp Ile Gly Ile Gly Ser Val Gly
20 25 30
Val Gly Ile Leu Asn Lys Val Thr Gly Glu Ile Ile His Lys Asn Ser
35 40 45
Arg Ile Phe Pro Ala Ala Gln Ala Glu Asn Asn Leu Val Arg Arg Thr
50 55 60
Asn Arg Gln Gly Arg Arg Leu Ala Arg Arg Lys Lys His Arg Arg Val
65 70 75 80
Arg Leu Asn Arg Leu Phe Glu Glu Ser Gly Leu Ile Thr Asp Phe Thr
85 90 95
Lys Ile Ser Ile Asn Leu Asn Pro Tyr Gln Leu Arg Val Lys Gly Leu
100 105 110
Thr Asp Glu Leu Ser Asn Glu Glu Leu Phe Ile Ala Leu Lys Asn Met
115 120 125
Val Lys His Arg Gly Ile Ser Tyr Leu Asp Asp Ala Ser Asp Asp Gly
130 135 140
Asn Ser Ser Val Gly Asp Tyr Ala Gln Ile Val Lys Glu Asn Ser Lys
145 150 155 160
Gln Leu Glu Thr Lys Thr Pro Gly Gln Ile Gln Leu Glu Arg Tyr Gln
165 170 175
Thr Tyr Gly Gln Leu Arg Gly Asp Phe Thr Val Glu Lys Asp Gly Lys
180 185 190
Lys His Arg Leu Ile Asn Val Phe Pro Thr Ser Ala Tyr Arg Ser Glu
195 200 205
Ala Leu Arg Ile Leu Gln Thr Gln Gln Glu Phe Asn Pro Gln Ile Thr
210 215 220
Asp Glu Phe Ile Asn Arg Tyr Leu Glu Ile Leu Thr Gly Lys Arg Lys
225 230 235 240
Tyr Tyr His Gly Pro Gly Asn Glu Lys Ser Arg Thr Asp Tyr Gly Arg
245 250 255
Tyr Arg Thr Ser Gly Glu Thr Leu Asp Asn Ile Phe Gly Ile Leu Ile
260 265 270
Gly Lys Cys Thr Phe Tyr Pro Asp Glu Phe Arg Ala Ala Lys Ala Ser
275 280 285
Tyr Thr Ala Gln Glu Phe Asn Leu Leu Asn Asp Leu Asn Asn Leu Thr
290 295 300
Val Pro Thr Glu Thr Lys Lys Leu Ser Lys Glu Gln Lys Asn Gln Ile
305 310 315 320
Ile Asn Tyr Val Lys Asn Glu Lys Ala Met Gly Pro Ala Lys Leu Phe
325 330 335
Lys Tyr Ile Ala Lys Leu Leu Ser Cys Asp Val Ala Asp Ile Lys Gly
340 345 350
Tyr Arg Ile Asp Lys Ser Gly Lys Ala Glu Ile His Thr Phe Glu Ala
355 360 365
Tyr Arg Lys Met Lys Thr Leu Glu Thr Leu Asp Ile Glu Gln Met Asp
370 375 380
Arg Glu Thr Leu Asp Lys Leu Ala Tyr Val Leu Thr Leu Asn Thr Glu
385 390 395 400
Arg Glu Gly Ile Gln Glu Ala Leu Glu His Glu Phe Ala Asp Gly Ser
405 410 415
Phe Ser Gln Lys Gln Val Asp Glu Leu Val Gln Phe Arg Lys Ala Asn
420 425 430
Ser Ser Ile Phe Gly Lys Gly Trp His Asn Phe Ser Val Lys Leu Met
435 440 445
Met Glu Leu Ile Pro Glu Leu Tyr Glu Thr Ser Glu Glu Gln Met Thr
450 455 460
Ile Leu Thr Arg Leu Gly Lys Gln Lys Thr Thr Ser Ser Ser Asn Lys
465 470 475 480
Thr Lys Tyr Ile Asp Glu Lys Leu Leu Thr Glu Glu Ile Tyr Asn Pro
485 490 495
Val Val Ala Lys Ser Val Arg Gln Ala Ile Lys Ile Val Asn Ala Ala
500 505 510
Ile Lys Glu Tyr Gly Asp Phe Asp Asn Ile Val Ile Glu Met Ala Arg
515 520 525
Glu Thr Asn Glu Asp Asp Glu Lys Lys Ala Ile Gln Lys Ile Gln Lys
530 535 540
Ala Asn Lys Asp Glu Lys Asp Ala Ala Met Leu Lys Ala Ala Asn Gln
545 550 555 560
Tyr Asn Gly Lys Ala Glu Leu Pro His Ser Val Phe His Gly His Lys
565 570 575
Gln Leu Ala Thr Lys Ile Arg Leu Trp His Gln Gln Gly Glu Arg Cys
580 585 590
Leu Tyr Thr Gly Lys Thr Ile Ser Ile His Asp Leu Ile Asn Asn Ser
595 600 605
Asn Gln Phe Glu Val Asp His Ile Leu Pro Leu Ser Ile Thr Phe Asp
610 615 620
Asp Ser Leu Ala Asn Lys Val Leu Val Tyr Ala Thr Ala Asn Gln Glu
625 630 635 640
Lys Gly Gln Arg Thr Pro Tyr Gln Ala Leu Asp Ser Met Asp Asp Ala
645 650 655
Trp Ser Phe Arg Glu Leu Lys Ala Phe Val Arg Glu Ser Lys Thr Leu
660 665 670
Ser Asn Lys Lys Lys Glu Tyr Leu Leu Thr Glu Glu Asp Ile Ser Lys
675 680 685
Phe Asp Val Arg Lys Lys Phe Ile Glu Arg Asn Leu Val Asp Thr Arg
690 695 700
Tyr Ala Ser Arg Val Val Leu Asn Ala Leu Gln Glu His Phe Arg Ala
705 710 715 720
His Lys Ile Asp Thr Lys Val Ser Val Val Arg Gly Gln Phe Thr Ser
725 730 735
Gln Leu Arg Arg His Trp Gly Ile Glu Lys Thr Arg Asp Thr Tyr His
740 745 750
His His Ala Val Asp Ala Leu Ile Ile Ala Ala Ser Ser Gln Leu Asn
755 760 765
Leu Trp Lys Lys Gln Lys Asn Thr Leu Val Ser Tyr Ser Glu Asp Gln
770 775 780
Leu Leu Asp Ile Glu Thr Gly Glu Leu Ile Ser Asp Asp Glu Tyr Lys
785 790 795 800
Glu Ser Val Phe Lys Ala Pro Tyr Gln His Phe Val Asp Thr Leu Lys
805 810 815
Ser Lys Glu Phe Glu Asp Ser Ile Leu Phe Ser Tyr Gln Val Asp Ser
820 825 830
Lys Phe Asn Arg Lys Ile Ser Asp Ala Thr Ile Tyr Ala Thr Arg Gln
835 840 845
Ala Lys Val Gly Lys Asp Lys Ala Asp Glu Thr Tyr Val Leu Gly Lys
850 855 860
Ile Lys Asp Ile Tyr Thr Gln Asp Gly Tyr Asp Ala Phe Met Lys Ile
865 870 875 880
Tyr Lys Lys Asp Lys Ser Lys Phe Leu Met Tyr Arg His Asp Pro Gln
885 890 895
Thr Phe Glu Lys Val Ile Glu Pro Ile Leu Glu Asn Tyr Pro Asn Lys
900 905 910
Gln Ile Asn Glu Lys Gly Lys Glu Val Pro Cys Asn Pro Phe Leu Lys
915 920 925
Tyr Lys Glu Glu His Gly Tyr Ile Arg Lys Tyr Ser Lys Lys Gly Asn
930 935 940
Gly Pro Glu Ile Lys Ser Leu Lys Tyr Tyr Asp Ser Lys Leu Gly Asn
945 950 955 960
His Ile Asp Ile Thr Pro Lys Asp Ser Asn Asn Lys Val Val Leu Gln
965 970 975
Ser Val Ser Pro Trp Arg Ala Asp Val Tyr Phe Asn Lys Thr Thr Gly
980 985 990
Lys Tyr Glu Ile Leu Gly Leu Lys Tyr Ala Asp Leu Gln Phe Glu Lys
995 1000 1005
Gly Thr Gly Thr Tyr Lys Ile Ser Gln Glu Lys Tyr Asn Asp Ile
1010 1015 1020
Lys Lys Lys Glu Gly Val Asp Ser Asp Ser Glu Phe Lys Phe Thr
1025 1030 1035
Leu Tyr Lys Asn Asp Leu Leu Leu Val Lys Asp Thr Glu Thr Lys
1040 1045 1050
Glu Gln Gln Leu Phe Arg Phe Leu Ser Arg Thr Met Pro Lys Gln
1055 1060 1065
Lys His Tyr Val Glu Leu Lys Pro Tyr Asp Lys Gln Lys Phe Glu
1070 1075 1080
Gly Gly Glu Ala Leu Ile Lys Val Leu Gly Asn Val Ala Asn Ser
1085 1090 1095
Gly Gln Cys Lys Lys Gly Leu Gly Lys Ser Asn Ile Ser Ile Tyr
1100 1105 1110
Lys Val Arg Thr Asp Val Leu Gly Asn Gln His Ile Ile Lys Asn
1115 1120 1125
Glu Gly Asp Lys Pro Lys Leu Asp Phe Lys Arg Pro Ala Ala Thr
1130 1135 1140
Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys
1145 1150
<210> 52
<211> 340
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 52
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccg ttacttaaat cttgcagaag ctacaaagat aaggcttcat gccgaaatca 300
acaccctgtc attttatggc agggtgtttt cgttatttaa 340
<210> 53
<211> 360
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (288)..(317)
<223> a, c, t, g, unknown or other
<400> 53
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccg ggttttagag ctatgctgtt ttgaatggtc ccaaaacnnn nnnnnnnnnn 300
nnnnnnnnnn nnnnnnngtt ttagagctat gctgttttga atggtcccaa aacttttttt 360
<210> 54
<211> 318
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (250)..(269)
<223> a, c, t, g, unknown or other
<400> 54
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccn nnnnnnnnnn nnnnnnnnng ttttagagct agaaatagca agttaaaata 300
aggctagtcc gttttttt 318
<210> 55
<211> 325
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (250)..(269)
<223> a, c, t, g, unknown or other
<400> 55
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccn nnnnnnnnnn nnnnnnnnng ttttagagct agaaatagca agttaaaata 300
aggctagtcc gttatcattt ttttt 325
<210> 56
<211> 337
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (250)..(269)
<223> a, c, t, g, unknown or other
<400> 56
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccn nnnnnnnnnn nnnnnnnnng ttttagagct agaaatagca agttaaaata 300
aggctagtcc gttatcaact tgaaaaagtg ttttttt 337
<210> 57
<211> 352
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (250)..(269)
<223> a, c, t, g, unknown or other
<400> 57
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccn nnnnnnnnnn nnnnnnnnng ttttagagct agaaatagca agttaaaata 300
aggctagtcc gttatcaact tgaaaaagtg gcaccgagtc ggtgcttttt tt 352
<210> 58
<211> 5101
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 58
cgttacataa cttacggtaa atggcccgcc tggctgaccg cccaacgacc cccgcccatt 60
gacgtcaata atgacgtatg ttcccatagt aacgccaata gggactttcc attgacgtca 120
atgggtggag tatttacggt aaactgccca cttggcagta catcaagtgt atcatatgcc 180
aagtacgccc cctattgacg tcaatgacgg taaatggccc gcctggcatt atgcccagta 240
catgacctta tgggactttc ctacttggca gtacatctac gtattagtca tcgctattac 300
catggtcgag gtgagcccca cgttctgctt cactctcccc atctcccccc cctccccacc 360
cccaattttg tatttattta ttttttaatt attttgtgca gcgatggggg cggggggggg 420
gggggggcgc gcgccaggcg gggcggggcg gggcgagggg cggggcgggg cgaggcggag 480
aggtgcggcg gcagccaatc agagcggcgc gctccgaaag tttcctttta tggcgaggcg 540
gcggcggcgg cggccctata aaaagcgaag cgcgcggcgg gcgggagtcg ctgcgacgct 600
gccttcgccc cgtgccccgc tccgccgccg cctcgcgccg cccgccccgg ctctgactga 660
ccgcgttact cccacaggtg agcgggcggg acggcccttc tcctccgggc tgtaattagc 720
tgagcaagag gtaagggttt aagggatggt tggttggtgg ggtattaatg tttaattacc 780
tggagcacct gcctgaaatc actttttttc aggttggacc ggtgccacca tggactataa 840
ggaccacgac ggagactaca aggatcatga tattgattac aaagacgatg acgataagat 900
ggccccaaag aagaagcgga aggtcggtat ccacggagtc ccagcagccg acaagaagta 960
cagcatcggc ctggacatcg gcaccaactc tgtgggctgg gccgtgatca ccgacgagta 1020
caaggtgccc agcaagaaat tcaaggtgct gggcaacacc gaccggcaca gcatcaagaa 1080
gaacctgatc ggagccctgc tgttcgacag cggcgaaaca gccgaggcca cccggctgaa 1140
gagaaccgcc agaagaagat acaccagacg gaagaaccgg atctgctatc tgcaagagat 1200
cttcagcaac gagatggcca aggtggacga cagcttcttc cacagactgg aagagtcctt 1260
cctggtggaa gaggataaga agcacgagcg gcaccccatc ttcggcaaca tcgtggacga 1320
ggtggcctac cacgagaagt accccaccat ctaccacctg agaaagaaac tggtggacag 1380
caccgacaag gccgacctgc ggctgatcta tctggccctg gcccacatga tcaagttccg 1440
gggccacttc ctgatcgagg gcgacctgaa ccccgacaac agcgacgtgg acaagctgtt 1500
catccagctg gtgcagacct acaaccagct gttcgaggaa aaccccatca acgccagcgg 1560
cgtggacgcc aaggccatcc tgtctgccag actgagcaag agcagacggc tggaaaatct 1620
gatcgcccag ctgcccggcg agaagaagaa tggcctgttc ggcaacctga ttgccctgag 1680
cctgggcctg acccccaact tcaagagcaa cttcgacctg gccgaggatg ccaaactgca 1740
gctgagcaag gacacctacg acgacgacct ggacaacctg ctggcccaga tcggcgacca 1800
gtacgccgac ctgtttctgg ccgccaagaa cctgtccgac gccatcctgc tgagcgacat 1860
cctgagagtg aacaccgaga tcaccaaggc ccccctgagc gcctctatga tcaagagata 1920
cgacgagcac caccaggacc tgaccctgct gaaagctctc gtgcggcagc agctgcctga 1980
gaagtacaaa gagattttct tcgaccagag caagaacggc tacgccggct acattgacgg 2040
cggagccagc caggaagagt tctacaagtt catcaagccc atcctggaaa agatggacgg 2100
caccgaggaa ctgctcgtga agctgaacag agaggacctg ctgcggaagc agcggacctt 2160
cgacaacggc agcatccccc accagatcca cctgggagag ctgcacgcca ttctgcggcg 2220
gcaggaagat ttttacccat tcctgaagga caaccgggaa aagatcgaga agatcctgac 2280
cttccgcatc ccctactacg tgggccctct ggccagggga aacagcagat tcgcctggat 2340
gaccagaaag agcgaggaaa ccatcacccc ctggaacttc gaggaagtgg tggacaaggg 2400
cgcttccgcc cagagcttca tcgagcggat gaccaacttc gataagaacc tgcccaacga 2460
gaaggtgctg cccaagcaca gcctgctgta cgagtacttc accgtgtata acgagctgac 2520
caaagtgaaa tacgtgaccg agggaatgag aaagcccgcc ttcctgagcg gcgagcagaa 2580
aaaggccatc gtggacctgc tgttcaagac caaccggaaa gtgaccgtga agcagctgaa 2640
agaggactac ttcaagaaaa tcgagtgctt cgactccgtg gaaatctccg gcgtggaaga 2700
tcggttcaac gcctccctgg gcacatacca cgatctgctg aaaattatca aggacaagga 2760
cttcctggac aatgaggaaa acgaggacat tctggaagat atcgtgctga ccctgacact 2820
gtttgaggac agagagatga tcgaggaacg gctgaaaacc tatgcccacc tgttcgacga 2880
caaagtgatg aagcagctga agcggcggag atacaccggc tggggcaggc tgagccggaa 2940
gctgatcaac ggcatccggg acaagcagtc cggcaagaca atcctggatt tcctgaagtc 3000
cgacggcttc gccaacagaa acttcatgca gctgatccac gacgacagcc tgacctttaa 3060
agaggacatc cagaaagccc aggtgtccgg ccagggcgat agcctgcacg agcacattgc 3120
caatctggcc ggcagccccg ccattaagaa gggcatcctg cagacagtga aggtggtgga 3180
cgagctcgtg aaagtgatgg gccggcacaa gcccgagaac atcgtgatcg aaatggccag 3240
agagaaccag accacccaga agggacagaa gaacagccgc gagagaatga agcggatcga 3300
agagggcatc aaagagctgg gcagccagat cctgaaagaa caccccgtgg aaaacaccca 3360
gctgcagaac gagaagctgt acctgtacta cctgcagaat gggcgggata tgtacgtgga 3420
ccaggaactg gacatcaacc ggctgtccga ctacgatgtg gaccatatcg tgcctcagag 3480
ctttctgaag gacgactcca tcgacaacaa ggtgctgacc agaagcgaca agaaccgggg 3540
caagagcgac aacgtgccct ccgaagaggt cgtgaagaag atgaagaact actggcggca 3600
gctgctgaac gccaagctga ttacccagag aaagttcgac aatctgacca aggccgagag 3660
aggcggcctg agcgaactgg ataaggccgg cttcatcaag agacagctgg tggaaacccg 3720
gcagatcaca aagcacgtgg cacagatcct ggactcccgg atgaacacta agtacgacga 3780
gaatgacaag ctgatccggg aagtgaaagt gatcaccctg aagtccaagc tggtgtccga 3840
tttccggaag gatttccagt tttacaaagt gcgcgagatc aacaactacc accacgccca 3900
cgacgcctac ctgaacgccg tcgtgggaac cgccctgatc aaaaagtacc ctaagctgga 3960
aagcgagttc gtgtacggcg actacaaggt gtacgacgtg cggaagatga tcgccaagag 4020
cgagcaggaa atcggcaagg ctaccgccaa gtacttcttc tacagcaaca tcatgaactt 4080
tttcaagacc gagattaccc tggccaacgg cgagatccgg aagcggcctc tgatcgagac 4140
aaacggcgaa accggggaga tcgtgtggga taagggccgg gattttgcca ccgtgcggaa 4200
agtgctgagc atgccccaag tgaatatcgt gaaaaagacc gaggtgcaga caggcggctt 4260
cagcaaagag tctatcctgc ccaagaggaa cagcgataag ctgatcgcca gaaagaagga 4320
ctgggaccct aagaagtacg gcggcttcga cagccccacc gtggcctatt ctgtgctggt 4380
ggtggccaaa gtggaaaagg gcaagtccaa gaaactgaag agtgtgaaag agctgctggg 4440
gatcaccatc atggaaagaa gcagcttcga gaagaatccc atcgactttc tggaagccaa 4500
gggctacaaa gaagtgaaaa aggacctgat catcaagctg cctaagtact ccctgttcga 4560
gctggaaaac ggccggaaga gaatgctggc ctctgccggc gaactgcaga agggaaacga 4620
actggccctg ccctccaaat atgtgaactt cctgtacctg gccagccact atgagaagct 4680
gaagggctcc cccgaggata atgagcagaa acagctgttt gtggaacagc acaagcacta 4740
cctggacgag atcatcgagc agatcagcga gttctccaag agagtgatcc tggccgacgc 4800
taatctggac aaagtgctgt ccgcctacaa caagcaccgg gataagccca tcagagagca 4860
ggccgagaat atcatccacc tgtttaccct gaccaatctg ggagcccctg ccgccttcaa 4920
gtactttgac accaccatcg accggaagag gtacaccagc accaaagagg tgctggacgc 4980
caccctgatc caccagagca tcaccggcct gtacgagaca cggatcgacc tgtctcagct 5040
gggaggcgac tttctttttc ttagcttgac cagctttctt agtagcagca ggacgcttta 5100
a 5101
<210> 59
<211> 137
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 59
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatt tagaaataaa tcttgcagaa 60
gctacaaaga taaggcttca tgccgaaatc aacaccctgt cattttatgg cagggtgttt 120
tcgttattta atttttt 137
<210> 60
<211> 123
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 60
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcagaaat gcagaagcta caaagataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttcgt tatttaattt 120
ttt 123
<210> 61
<211> 110
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 61
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcagaaat gcagaagcta caaagataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttttt 110
<210> 62
<211> 137
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 62
nnnnnnnnnn nnnnnnnnnn gttattgtac tctcaagatt tagaaataaa tcttgcagaa 60
gctacaaaga taaggcttca tgccgaaatc aacaccctgt cattttatgg cagggtgttt 120
tcgttattta atttttt 137
<210> 63
<211> 123
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 63
nnnnnnnnnn nnnnnnnnnn gttattgtac tctcagaaat gcagaagcta caaagataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttcgt tatttaattt 120
ttt 123
<210> 64
<211> 110
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 64
nnnnnnnnnn nnnnnnnnnn gttattgtac tctcagaaat gcagaagcta caaagataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttttt 110
<210> 65
<211> 137
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 65
nnnnnnnnnn nnnnnnnnnn gttattgtac tctcaagatt tagaaataaa tcttgcagaa 60
gctacaatga taaggcttca tgccgaaatc aacaccctgt cattttatgg cagggtgttt 120
tcgttattta atttttt 137
<210> 66
<211> 123
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 66
nnnnnnnnnn nnnnnnnnnn gttattgtac tctcagaaat gcagaagcta caatgataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttcgt tatttaattt 120
ttt 123
<210> 67
<211> 110
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 67
nnnnnnnnnn nnnnnnnnnn gttattgtac tctcagaaat gcagaagcta caatgataag 60
gcttcatgcc gaaatcaaca ccctgtcatt ttatggcagg gtgttttttt 110
<210> 68
<211> 107
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 68
nnnnnnnnnn nnnnnnnnnn gttttagagc tgtggaaaca cagcgagtta aaataaggct 60
tagtccgtac tcaacttgaa aaggtggcac cgattcggtg ttttttt 107
<210> 69
<211> 4263
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 69
atgaaaaggc cggcggccac gaaaaaggcc ggccaggcaa aaaagaaaaa gaccaagccc 60
tacagcatcg gcctggacat cggcaccaat agcgtgggct gggccgtgac caccgacaac 120
tacaaggtgc ccagcaagaa aatgaaggtg ctgggcaaca cctccaagaa gtacatcaag 180
aaaaacctgc tgggcgtgct gctgttcgac agcggcatta cagccgaggg cagacggctg 240
aagagaaccg ccagacggcg gtacacccgg cggagaaaca gaatcctgta tctgcaagag 300
atcttcagca ccgagatggc taccctggac gacgccttct tccagcggct ggacgacagc 360
ttcctggtgc ccgacgacaa gcgggacagc aagtacccca tcttcggcaa cctggtggaa 420
gagaaggcct accacgacga gttccccacc atctaccacc tgagaaagta cctggccgac 480
agcaccaaga aggccgacct gagactggtg tatctggccc tggcccacat gatcaagtac 540
cggggccact tcctgatcga gggcgagttc aacagcaaga acaacgacat ccagaagaac 600
ttccaggact tcctggacac ctacaacgcc atcttcgaga gcgacctgtc cctggaaaac 660
agcaagcagc tggaagagat cgtgaaggac aagatcagca agctggaaaa gaaggaccgc 720
atcctgaagc tgttccccgg cgagaagaac agcggaatct tcagcgagtt tctgaagctg 780
atcgtgggca accaggccga cttcagaaag tgcttcaacc tggacgagaa agccagcctg 840
cacttcagca aagagagcta cgacgaggac ctggaaaccc tgctgggata tatcggcgac 900
gactacagcg acgtgttcct gaaggccaag aagctgtacg acgctatcct gctgagcggc 960
ttcctgaccg tgaccgacaa cgagacagag gccccactga gcagcgccat gattaagcgg 1020
tacaacgagc acaaagagga tctggctctg ctgaaagagt acatccggaa catcagcctg 1080
aaaacctaca atgaggtgtt caaggacgac accaagaacg gctacgccgg ctacatcgac 1140
ggcaagacca accaggaaga tttctatgtg tacctgaaga agctgctggc cgagttcgag 1200
ggggccgact actttctgga aaaaatcgac cgcgaggatt tcctgcggaa gcagcggacc 1260
ttcgacaacg gcagcatccc ctaccagatc catctgcagg aaatgcgggc catcctggac 1320
aagcaggcca agttctaccc attcctggcc aagaacaaag agcggatcga gaagatcctg 1380
accttccgca tcccttacta cgtgggcccc ctggccagag gcaacagcga ttttgcctgg 1440
tccatccgga agcgcaatga gaagatcacc ccctggaact tcgaggacgt gatcgacaaa 1500
gagtccagcg ccgaggcctt catcaaccgg atgaccagct tcgacctgta cctgcccgag 1560
gaaaaggtgc tgcccaagca cagcctgctg tacgagacat tcaatgtgta taacgagctg 1620
accaaagtgc ggtttatcgc cgagtctatg cgggactacc agttcctgga ctccaagcag 1680
aaaaaggaca tcgtgcggct gtacttcaag gacaagcgga aagtgaccga taaggacatc 1740
atcgagtacc tgcacgccat ctacggctac gatggcatcg agctgaaggg catcgagaag 1800
cagttcaact ccagcctgag cacataccac gacctgctga acattatcaa cgacaaagaa 1860
tttctggacg actccagcaa cgaggccatc atcgaagaga tcatccacac cctgaccatc 1920
tttgaggacc gcgagatgat caagcagcgg ctgagcaagt tcgagaacat cttcgacaag 1980
agcgtgctga aaaagctgag cagacggcac tacaccggct ggggcaagct gagcgccaag 2040
ctgatcaacg gcatccggga cgagaagtcc ggcaacacaa tcctggacta cctgatcgac 2100
gacggcatca gcaaccggaa cttcatgcag ctgatccacg acgacgccct gagcttcaag 2160
aagaagatcc agaaggccca gatcatcggg gacgaggaca agggcaacat caaagaagtc 2220
gtgaagtccc tgcccggcag ccccgccatc aagaagggaa tcctgcagag catcaagatc 2280
gtggacgagc tcgtgaaagt gatgggcggc agaaagcccg agagcatcgt ggtggaaatg 2340
gctagagaga accagtacac caatcagggc aagagcaaca gccagcagag actgaagaga 2400
ctggaaaagt ccctgaaaga gctgggcagc aagattctga aagagaatat ccctgccaag 2460
ctgtccaaga tcgacaacaa cgccctgcag aacgaccggc tgtacctgta ctacctgcag 2520
aatggcaagg acatgtatac aggcgacgac ctggatatcg accgcctgag caactacgac 2580
atcgaccata ttatccccca ggccttcctg aaagacaaca gcattgacaa caaagtgctg 2640
gtgtcctccg ccagcaaccg cggcaagtcc gatgatgtgc ccagcctgga agtcgtgaaa 2700
aagagaaaga ccttctggta tcagctgctg aaaagcaagc tgattagcca gaggaagttc 2760
gacaacctga ccaaggccga gagaggcggc ctgagccctg aagataaggc cggcttcatc 2820
cagagacagc tggtggaaac ccggcagatc accaagcacg tggccagact gctggatgag 2880
aagtttaaca acaagaagga cgagaacaac cgggccgtgc ggaccgtgaa gatcatcacc 2940
ctgaagtcca ccctggtgtc ccagttccgg aaggacttcg agctgtataa agtgcgcgag 3000
atcaatgact ttcaccacgc ccacgacgcc tacctgaatg ccgtggtggc ttccgccctg 3060
ctgaagaagt accctaagct ggaacccgag ttcgtgtacg gcgactaccc caagtacaac 3120
tccttcagag agcggaagtc cgccaccgag aaggtgtact tctactccaa catcatgaat 3180
atctttaaga agtccatctc cctggccgat ggcagagtga tcgagcggcc cctgatcgaa 3240
gtgaacgaag agacaggcga gagcgtgtgg aacaaagaaa gcgacctggc caccgtgcgg 3300
cgggtgctga gttatcctca agtgaatgtc gtgaagaagg tggaagaaca gaaccacggc 3360
ctggatcggg gcaagcccaa gggcctgttc aacgccaacc tgtccagcaa gcctaagccc 3420
aactccaacg agaatctcgt gggggccaaa gagtacctgg accctaagaa gtacggcgga 3480
tacgccggca tctccaatag cttcaccgtg ctcgtgaagg gcacaatcga gaagggcgct 3540
aagaaaaaga tcacaaacgt gctggaattt caggggatct ctatcctgga ccggatcaac 3600
taccggaagg ataagctgaa ctttctgctg gaaaaaggct acaaggacat tgagctgatt 3660
atcgagctgc ctaagtactc cctgttcgaa ctgagcgacg gctccagacg gatgctggcc 3720
tccatcctgt ccaccaacaa caagcggggc gagatccaca agggaaacca gatcttcctg 3780
agccagaaat ttgtgaaact gctgtaccac gccaagcgga tctccaacac catcaatgag 3840
aaccaccgga aatacgtgga aaaccacaag aaagagtttg aggaactgtt ctactacatc 3900
ctggagttca acgagaacta tgtgggagcc aagaagaacg gcaaactgct gaactccgcc 3960
ttccagagct ggcagaacca cagcatcgac gagctgtgca gctccttcat cggccctacc 4020
ggcagcgagc ggaagggact gtttgagctg acctccagag gctctgccgc cgactttgag 4080
ttcctgggag tgaagatccc ccggtacaga gactacaccc cctctagtct gctgaaggac 4140
gccaccctga tccaccagag cgtgaccggc ctgtacgaaa cccggatcga cctggctaag 4200
ctgggcgagg gaaagcgtcc tgctgctact aagaaagctg gtcaagctaa gaaaaagaaa 4260
taa 4263
<210> 70
<211> 84
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 70
ggaaccattc ataacagcat agcaagttat aataaggcta gtccgttatc aacttgaaaa 60
agtggcaccg agtcggtgct tttt 84
<210> 71
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 71
gttatagagc tatgctgtta tgaatggtcc caaaac 36
<210> 72
<211> 84
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 72
ggaaccattc aatacagcat agcaagttaa tataaggcta gtccgttatc aacttgaaaa 60
agtggcaccg agtcggtgct tttt 84
<210> 73
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 73
gtattagagc tatgctgtat tgaatggtcc caaaac 36
<210> 74
<211> 103
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 74
nnnnnnnnnn nnnnnnnnnn gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcaac ttgaaaaagt ggcaccgagt cggtgctttt ttt 103
<210> 75
<211> 103
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 75
nnnnnnnnnn nnnnnnnnnn gtattagagc tagaaatagc aagttaatat aaggctagtc 60
cgttatcaac ttgaaaaagt ggcaccgagt cggtgctttt ttt 103
<210> 76
<211> 123
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 76
nnnnnnnnnn nnnnnnnnnn gttttagagc tatgctgttt tggaaacaaa acagcatagc 60
aagttaaaat aaggctagtc cgttatcaac ttgaaaaagt ggcaccgagt cggtgctttt 120
ttt 123
<210> 77
<211> 123
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 77
nnnnnnnnnn nnnnnnnnnn gtattagagc tatgctgtat tggaaacaat acagcatagc 60
aagttaatat aaggctagtc cgttatcaac ttgaaaaagt ggcaccgagt cggtgctttt 120
ttt 123
<210> 78
<211> 20
<212> DNA
<213> Homo sapiens
<400> 78
gtcacctcca atgactaggg 20
<210> 79
<211> 984
<212> PRT
<213> Campylobacter jejuni
<400> 79
Met Ala Arg Ile Leu Ala Phe Asp Ile Gly Ile Ser Ser Ile Gly Trp
1 5 10 15
Ala Phe Ser Glu Asn Asp Glu Leu Lys Asp Cys Gly Val Arg Ile Phe
20 25 30
Thr Lys Val Glu Asn Pro Lys Thr Gly Glu Ser Leu Ala Leu Pro Arg
35 40 45
Arg Leu Ala Arg Ser Ala Arg Lys Arg Leu Ala Arg Arg Lys Ala Arg
50 55 60
Leu Asn His Leu Lys His Leu Ile Ala Asn Glu Phe Lys Leu Asn Tyr
65 70 75 80
Glu Asp Tyr Gln Ser Phe Asp Glu Ser Leu Ala Lys Ala Tyr Lys Gly
85 90 95
Ser Leu Ile Ser Pro Tyr Glu Leu Arg Phe Arg Ala Leu Asn Glu Leu
100 105 110
Leu Ser Lys Gln Asp Phe Ala Arg Val Ile Leu His Ile Ala Lys Arg
115 120 125
Arg Gly Tyr Asp Asp Ile Lys Asn Ser Asp Asp Lys Glu Lys Gly Ala
130 135 140
Ile Leu Lys Ala Ile Lys Gln Asn Glu Glu Lys Leu Ala Asn Tyr Gln
145 150 155 160
Ser Val Gly Glu Tyr Leu Tyr Lys Glu Tyr Phe Gln Lys Phe Lys Glu
165 170 175
Asn Ser Lys Glu Phe Thr Asn Val Arg Asn Lys Lys Glu Ser Tyr Glu
180 185 190
Arg Cys Ile Ala Gln Ser Phe Leu Lys Asp Glu Leu Lys Leu Ile Phe
195 200 205
Lys Lys Gln Arg Glu Phe Gly Phe Ser Phe Ser Lys Lys Phe Glu Glu
210 215 220
Glu Val Leu Ser Val Ala Phe Tyr Lys Arg Ala Leu Lys Asp Phe Ser
225 230 235 240
His Leu Val Gly Asn Cys Ser Phe Phe Thr Asp Glu Lys Arg Ala Pro
245 250 255
Lys Asn Ser Pro Leu Ala Phe Met Phe Val Ala Leu Thr Arg Ile Ile
260 265 270
Asn Leu Leu Asn Asn Leu Lys Asn Thr Glu Gly Ile Leu Tyr Thr Lys
275 280 285
Asp Asp Leu Asn Ala Leu Leu Asn Glu Val Leu Lys Asn Gly Thr Leu
290 295 300
Thr Tyr Lys Gln Thr Lys Lys Leu Leu Gly Leu Ser Asp Asp Tyr Glu
305 310 315 320
Phe Lys Gly Glu Lys Gly Thr Tyr Phe Ile Glu Phe Lys Lys Tyr Lys
325 330 335
Glu Phe Ile Lys Ala Leu Gly Glu His Asn Leu Ser Gln Asp Asp Leu
340 345 350
Asn Glu Ile Ala Lys Asp Ile Thr Leu Ile Lys Asp Glu Ile Lys Leu
355 360 365
Lys Lys Ala Leu Ala Lys Tyr Asp Leu Asn Gln Asn Gln Ile Asp Ser
370 375 380
Leu Ser Lys Leu Glu Phe Lys Asp His Leu Asn Ile Ser Phe Lys Ala
385 390 395 400
Leu Lys Leu Val Thr Pro Leu Met Leu Glu Gly Lys Lys Tyr Asp Glu
405 410 415
Ala Cys Asn Glu Leu Asn Leu Lys Val Ala Ile Asn Glu Asp Lys Lys
420 425 430
Asp Phe Leu Pro Ala Phe Asn Glu Thr Tyr Tyr Lys Asp Glu Val Thr
435 440 445
Asn Pro Val Val Leu Arg Ala Ile Lys Glu Tyr Arg Lys Val Leu Asn
450 455 460
Ala Leu Leu Lys Lys Tyr Gly Lys Val His Lys Ile Asn Ile Glu Leu
465 470 475 480
Ala Arg Glu Val Gly Lys Asn His Ser Gln Arg Ala Lys Ile Glu Lys
485 490 495
Glu Gln Asn Glu Asn Tyr Lys Ala Lys Lys Asp Ala Glu Leu Glu Cys
500 505 510
Glu Lys Leu Gly Leu Lys Ile Asn Ser Lys Asn Ile Leu Lys Leu Arg
515 520 525
Leu Phe Lys Glu Gln Lys Glu Phe Cys Ala Tyr Ser Gly Glu Lys Ile
530 535 540
Lys Ile Ser Asp Leu Gln Asp Glu Lys Met Leu Glu Ile Asp His Ile
545 550 555 560
Tyr Pro Tyr Ser Arg Ser Phe Asp Asp Ser Tyr Met Asn Lys Val Leu
565 570 575
Val Phe Thr Lys Gln Asn Gln Glu Lys Leu Asn Gln Thr Pro Phe Glu
580 585 590
Ala Phe Gly Asn Asp Ser Ala Lys Trp Gln Lys Ile Glu Val Leu Ala
595 600 605
Lys Asn Leu Pro Thr Lys Lys Gln Lys Arg Ile Leu Asp Lys Asn Tyr
610 615 620
Lys Asp Lys Glu Gln Lys Asn Phe Lys Asp Arg Asn Leu Asn Asp Thr
625 630 635 640
Arg Tyr Ile Ala Arg Leu Val Leu Asn Tyr Thr Lys Asp Tyr Leu Asp
645 650 655
Phe Leu Pro Leu Ser Asp Asp Glu Asn Thr Lys Leu Asn Asp Thr Gln
660 665 670
Lys Gly Ser Lys Val His Val Glu Ala Lys Ser Gly Met Leu Thr Ser
675 680 685
Ala Leu Arg His Thr Trp Gly Phe Ser Ala Lys Asp Arg Asn Asn His
690 695 700
Leu His His Ala Ile Asp Ala Val Ile Ile Ala Tyr Ala Asn Asn Ser
705 710 715 720
Ile Val Lys Ala Phe Ser Asp Phe Lys Lys Glu Gln Glu Ser Asn Ser
725 730 735
Ala Glu Leu Tyr Ala Lys Lys Ile Ser Glu Leu Asp Tyr Lys Asn Lys
740 745 750
Arg Lys Phe Phe Glu Pro Phe Ser Gly Phe Arg Gln Lys Val Leu Asp
755 760 765
Lys Ile Asp Glu Ile Phe Val Ser Lys Pro Glu Arg Lys Lys Pro Ser
770 775 780
Gly Ala Leu His Glu Glu Thr Phe Arg Lys Glu Glu Glu Phe Tyr Gln
785 790 795 800
Ser Tyr Gly Gly Lys Glu Gly Val Leu Lys Ala Leu Glu Leu Gly Lys
805 810 815
Ile Arg Lys Val Asn Gly Lys Ile Val Lys Asn Gly Asp Met Phe Arg
820 825 830
Val Asp Ile Phe Lys His Lys Lys Thr Asn Lys Phe Tyr Ala Val Pro
835 840 845
Ile Tyr Thr Met Asp Phe Ala Leu Lys Val Leu Pro Asn Lys Ala Val
850 855 860
Ala Arg Ser Lys Lys Gly Glu Ile Lys Asp Trp Ile Leu Met Asp Glu
865 870 875 880
Asn Tyr Glu Phe Cys Phe Ser Leu Tyr Lys Asp Ser Leu Ile Leu Ile
885 890 895
Gln Thr Lys Asp Met Gln Glu Pro Glu Phe Val Tyr Tyr Asn Ala Phe
900 905 910
Thr Ser Ser Thr Val Ser Leu Ile Val Ser Lys His Asp Asn Lys Phe
915 920 925
Glu Thr Leu Ser Lys Asn Gln Lys Ile Leu Phe Lys Asn Ala Asn Glu
930 935 940
Lys Glu Val Ile Ala Lys Ser Ile Gly Ile Gln Asn Leu Lys Val Phe
945 950 955 960
Glu Lys Tyr Ile Val Ser Ala Leu Gly Glu Val Thr Lys Ala Glu Phe
965 970 975
Arg Gln Arg Glu Asp Phe Lys Lys
980
<210> 80
<211> 91
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 80
tataatctca taagaaattt aaaaagggac taaaataaag agtttgcggg actctgcggg 60
gttacaatcc cctaaaaccg cttttaaaat t 91
<210> 81
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 81
attttaccat aaagaaattt aaaaagggac taaaac 36
<210> 82
<211> 95
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 82
nnnnnnnnnn nnnnnnnnnn guuuuagucc cgaaagggac uaaaauaaag aguuugcggg 60
acucugcggg guuacaaucc ccuaaaaccg cuuuu 95
<210> 83
<211> 69
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 83
gucaccucca augacuaggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuuuuuu 69
<210> 84
<211> 69
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 84
gacaucgaug uccuccccau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuuuuuu 69
<210> 85
<211> 69
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 85
gaguccgagc agaagaagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuuuuuu 69
<210> 86
<211> 69
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 86
ggggccgaga uuggguguuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuuuuuu 69
<210> 87
<211> 69
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 87
guggcgagag gggccgagau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuuuuuu 69
<210> 88
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 88
gucaccucca augacuaggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucauu uuuuuu 76
<210> 89
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 89
gacaucgaug uccuccccau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucauu uuuuuu 76
<210> 90
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 90
gaguccgagc agaagaagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucauu uuuuuu 76
<210> 91
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 91
ggggccgaga uuggguguuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucauu uuuuuu 76
<210> 92
<211> 76
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 92
guggcgagag gggccgagau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucauu uuuuuu 76
<210> 93
<211> 88
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 93
gucaccucca augacuaggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu guuuuuuu 88
<210> 94
<211> 88
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 94
gacaucgaug uccuccccau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu guuuuuuu 88
<210> 95
<211> 88
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 95
gaguccgagc agaagaagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu guuuuuuu 88
<210> 96
<211> 88
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 96
ggggccgaga uuggguguuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu guuuuuuu 88
<210> 97
<211> 88
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 97
guggcgagag gggccgagau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu guuuuuuu 88
<210> 98
<211> 103
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 98
gucaccucca augacuaggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcuuuu uuu 103
<210> 99
<211> 103
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 99
gacaucgaug uccuccccau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcuuuu uuu 103
<210> 100
<211> 103
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 100
gaguccgagc agaagaagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcuuuu uuu 103
<210> 101
<211> 103
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 101
ggggccgaga uuggguguuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcuuuu uuu 103
<210> 102
<211> 103
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 102
guggcgagag gggccgagau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcuuuu uuu 103
<210> 103
<211> 102
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 103
gttttagagc tatgctgttt tgaatggtcc caaaacggaa gggcctgagt ccgagcagaa 60
gaagaagttt tagagctatg ctgttttgaa tggtcccaaa ac 102
<210> 104
<211> 100
<212> DNA
<213> Homo sapiens
<400> 104
cggaggacaa agtacaaacg gcagaagctg gaggaggaag ggcctgagtc cgagcagaag 60
aagaagggct cccatcacat caaccggtgg cgcattgcca 100
<210> 105
<211> 50
<212> DNA
<213> Homo sapiens
<400> 105
agctggagga ggaagggcct gagtccgagc agaagaagaa gggctcccac 50
<210> 106
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 106
gaguccgagc agaagaagaa guuuuagagc 30
<210> 107
<211> 49
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 107
agctggagga ggaagggcct gagtccgagc agaagagaag ggctcccat 49
<210> 108
<211> 53
<212> DNA
<213> Homo sapiens
<400> 108
ctggaggagg aagggcctga gtccgagcag aagaagaagg gctcccatca cat 53
<210> 109
<211> 52
<212> DNA
<213> Homo sapiens
<400> 109
ctggaggagg aagggcctga gtccgagcag aagagaaggg ctcccatcac at 52
<210> 110
<211> 54
<212> DNA
<213> Homo sapiens
<400> 110
ctggaggagg aagggcctga gtccgagcag aagaaagaag ggctcccatc acat 54
<210> 111
<211> 50
<212> DNA
<213> Homo sapiens
<400> 111
ctggaggagg aagggcctga gtccgagcag aagaagggct cccatcacat 50
<210> 112
<211> 47
<212> DNA
<213> Homo sapiens
<400> 112
ctggaggagg aagggcctga gcccgagcag aagggctccc atcacat 47
<210> 113
<211> 66
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> source
<223> /note="Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 113
nnnnnnnnnn nnnnnnnnnn guuuuagagc uagaaauagc aaguuaaaau aaggctagtc 60
cguuuu 66
<210> 114
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 114
gaguccgagc agaagaagaa 20
<210> 115
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 115
gacaucgaug uccuccccau 20
<210> 116
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 116
gucaccucca augacuaggg 20
<210> 117
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 117
auuggguguu cagggcagag 20
<210> 118
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 118
guggcgagag gggccgagau 20
<210> 119
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 119
ggggccgaga uuggguguuc 20
<210> 120
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 120
gugccauuag cuaaaugcau 20
<210> 121
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 121
guaccaccca caggugccag 20
<210> 122
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 122
gaaagccucu gggccaggaa 20
<210> 123
<211> 48
<212> DNA
<213> Homo sapiens
<400> 123
ctggaggagg aagggcctga gtccgagcag aagaagaagg gctcccat 48
<210> 124
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 124
gaguccgagc agaagaagau 20
<210> 125
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 125
gaguccgagc agaagaagua 20
<210> 126
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 126
gaguccgagc agaagaacaa 20
<210> 127
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 127
gaguccgagc agaagaugaa 20
<210> 128
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 128
gaguccgagc agaaguagaa 20
<210> 129
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 129
gaguccgagc agaugaagaa 20
<210> 130
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 130
gaguccgagc acaagaagaa 20
<210> 131
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 131
gaguccgagg agaagaagaa 20
<210> 132
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 132
gaguccgugc agaagaagaa 20
<210> 133
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 133
gagucggagc agaagaagaa 20
<210> 134
<211> 20
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 134
gagaccgagc agaagaagaa 20
<210> 135
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 135
aatgacaagc ttgctagcgg tggg 24
<210> 136
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 136
aaaacggaag ggcctgagtc cgagcagaag aagaagttt 39
<210> 137
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 137
aaacaggggc cgagattggg tgttcagggc agaggtttt 39
<210> 138
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 138
aaaacggaag ggcctgagtc cgagcagaag aagaagtt 38
<210> 139
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 139
aacggaggga ggggcacaga tgagaaactc agggttttag 40
<210> 140
<211> 38
<212> DNA
<213> Homo sapiens
<400> 140
agcccttctt cttctgctcg gactcaggcc cttcctcc 38
<210> 141
<211> 40
<212> DNA
<213> Homo sapiens
<400> 141
cagggaggga ggggcacaga tgagaaactc aggaggcccc 40
<210> 142
<211> 80
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 142
ggcaatgcgc caccggttga tgtgatggga gcccttctag gaggccccca gagcagccac 60
tggggcctca acactcaggc 80
<210> 143
<211> 98
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 143
ggacgaaaca ccggaaccat tcaaaacagc atagcaagtt aaaataaggc tagtccgtta 60
tcaacttgaa aaagtggcac cgagtcggtg cttttttt 98
<210> 144
<211> 186
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 144
ggacgaaaca ccggtagtat taagtattgt tttatggctg ataaatttct ttgaatttct 60
ccttgattat ttgttataaa agttataaaa taatcttgtt ggaaccattc aaaacagcat 120
agcaagttaa aataaggcta gtccgttatc aacttgaaaa agtggcaccg agtcggtgct 180
tttttt 186
<210> 145
<211> 46
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(19)
<223> a, c, u, g, unknown or other
<400> 145
nnnnnnnnnn nnnnnnnnng uuauuguacu cucaagauuu auuuuu 46
<210> 146
<211> 91
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 146
guuacuuaaa ucuugcagaa gcuacaaaga uaaggcuuca ugccgaaauc aacacccugu 60
cauuuuaugg caggguguuu ucguuauuua a 91
<210> 147
<211> 70
<212> DNA
<213> Homo sapiens
<400> 147
ttttctagtg ctgagtttct gtgactcctc tacattctac ttctctgtgt ttctgtatac 60
tacctcctcc 70
<210> 148
<211> 122
<212> DNA
<213> Homo sapiens
<400> 148
ggaggaaggg cctgagtccg agcagaagaa gaagggctcc catcacatca accggtggcg 60
cattgccacg aagcaggcca atggggagga catcgatgtc acctccaatg actagggtgg 120
gc 122
<210> 149
<211> 48
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (3)..(32)
<223> a, c, u, g, unknown or other
<400> 149
acnnnnnnnn nnnnnnnnnn nnnnnnnnnn nnguuuuaga gcuaugcu 48
<210> 150
<211> 67
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> source
<223> /note="Description of Combined DNA/RNA Molecule: Synthetic
oligonucleotide"
<400> 150
agcauagcaa guuaaaauaa ggctaguccg uuaucaacuu gaaaaagugg caccgagucg 60
gugcuuu 67
<210> 151
<211> 62
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 151
nnnnnnnnnn nnnnnnnnnn guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cg 62
<210> 152
<211> 73
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 152
tgaatggtcc caaaacggaa gggcctgagt ccgagcagaa gaagaagttt tagagctatg 60
ctgttttgaa tgg 73
<210> 153
<211> 99
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 153
nnnnnnnnnn nnnnnnnnnn guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcuuu 99
<210> 154
<211> 127
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 154
guuuuuguac ucucaagauu uaaguaacug uacaacguua cuuaaaucuu gcagaagcua 60
caaagauaag gcuucaugcc gaaaucaaca cccugucauu uuauggcagg guguuuucgu 120
uauuuaa 127
<210> 155
<211> 56
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 155
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatt taagtaactg tacaac 56
<210> 156
<211> 91
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 156
gttacttaaa tcttgcagaa gctacaaaga taaggcttca tgccgaaatc aacaccctgt 60
cattttatgg cagggtgttt tcgttattta a 91
<210> 157
<211> 134
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 157
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatt taaggaaact aaatcttgca 60
gaagctacaa agataaggct tcatgccgaa atcaacaccc tgtcatttta tggcagggtg 120
ttttcgttat ttaa 134
<210> 158
<211> 131
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 158
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatt tagaaataaa tcttgcagaa 60
gctacaaaga taaggcttca tgccgaaatc aacaccctgt cattttatgg cagggtgttt 120
tcgttattta a 131
<210> 159
<211> 125
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 159
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatg aaaatcttgc agaagctaca 60
aagataaggc ttcatgccga aatcaacacc ctgtcatttt atggcagggt gttttcgtta 120
tttaa 125
<210> 160
<211> 112
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 160
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctgaaaaga agctacaaag ataaggcttc 60
atgccgaaat caacaccctg tcattttatg gcagggtgtt ttcgttattt aa 112
<210> 161
<211> 107
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 161
nnnnnnnnnn nnnnnnnnnn gtttttgtac tgaaaagcta caaagataag gcttcatgcc 60
gaaatcaaca ccctgtcatt ttatggcagg gtgttttcgt tatttaa 107
<210> 162
<211> 108
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 162
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatt tagaaataaa tcttgcagaa 60
gctacaaaga taaggcttca tgccgaaatc aacaccctgt cattttat 108
<210> 163
<211> 86
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 163
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatt tagaaataaa tcttgcagaa 60
gctacaaaga taaggcttca tgccga 86
<210> 164
<211> 79
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 164
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatt tagaaataaa tcttgcagaa 60
gctacaaaga taaggcttc 79
<210> 165
<211> 73
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, t, g, unknown or other
<400> 165
nnnnnnnnnn nnnnnnnnnn gtttttgtac tctcaagatt tagaaataaa tcttgcagaa 60
gctacaaaga taa 73
<210> 166
<211> 125
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 166
guuuuagucc cuuuuuaaau uucuuuaugg uaaaauuaua aucucauaag aaauuuaaaa 60
agggacuaaa auaaagaguu ugcgggacuc ugcgggguua caauccccua aaaccgcuuu 120
uaaaa 125
<210> 167
<211> 91
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 167
guuacuuaaa ucuugcagaa gcuacaaaga uaaggcuuca ugccgaaauc aacacccugu 60
cauuuuaugg caggguguuu ucguuauuua a 91
<210> 168
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 168
gggacucaac caagucauuc guuuuuguac ucucaagauu uaaguaacug uacaac 56
<210> 169
<211> 147
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 169
gggacucaac caagucauuc guuuuuguac ucucaagauu uaaguaacug uacaacguua 60
cuuaaaucuu gcagaagcua caaagauaag gcuucaugcc gaaaucaaca cccugucauu 120
uuauggcagg guguuuucgu uauuuaa 147
<210> 170
<211> 70
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 170
cuugcagaag cuacaaagau aaggcuucau gccgaaauca acacccuguc auuuuauggc 60
aggguguuuu 70
<210> 171
<211> 42
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 171
gggacucaac caagucauuc guuuuuguac ucucaagauu ua 42
<210> 172
<211> 112
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 172
gggacucaac caagucauuc guuuuuguac ucucaagauu uacuugcaga agcuacaaag 60
auaaggcuuc augccgaaau caacacccug ucauuuuaug gcaggguguu uu 112
<210> 173
<211> 116
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 173
gggacucaac caagucauuc guuuuuguac ucucaagauu uagaaacuug cagaagcuac 60
aaagauaagg cuucaugccg aaaucaacac ccugucauuu uauggcaggg uguuuu 116
<210> 174
<211> 116
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 174
gggacucaac caagucauuc guuuuuguac ucucaagauu uagaaacuug cagaagcuac 60
aaagauaagg cuucaugccg aaaucaacac ccugucauuu uauggcaggg uguuuu 116
<210> 175
<211> 102
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 175
gggacucaac caagucauuc guuuuuguag aaauacaaag auaaggcuuc augccgaaau 60
caacacccug ucauuuuaug gcaggguguu uucguuauuu aa 102
<210> 176
<211> 102
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<400> 176
gggacucaac caagucauuc guuuuuguag aaauacaaag auaaggcuuc augccgaaau 60
caacacccug ucauuuuaug gcaggguguu uucguuauuu aa 102
<210> 177
<211> 57
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 177
gggacucaac caagucauuc guuuuuguag aaauacaaag auaaggcuuc augccga 57
<210> 178
<211> 57
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 178
gggacucaac caagucauuc guuuuuguag aaauacaaag auaaggcuuc augccga 57
<210> 179
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 179
gtggtgtcac gctcgtcgtt tgg 23
<210> 180
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<400> 180
tccagtctat taattgttgc cgg 23
<210> 181
<211> 64
<212> DNA
<213> Homo sapiens
<400> 181
caagaggctt gagtaggaga ggagtgccgc cgaggcgggg cggggcgggg cgtggagctg 60
ggct 64
<210> 182
<211> 99
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 182
nnnnnnnnnn nnnnnnnnnn guauuagagc uagaaauagc aaguuaauau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcuuu 99
<210> 183
<211> 119
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 183
nnnnnnnnnn nnnnnnnnnn guuuuagagc uaugcuguuu uggaaacaaa acagcauagc 60
aaguuaaaau aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugcuuu 119
<210> 184
<211> 119
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 184
nnnnnnnnnn nnnnnnnnnn guauuagagc uaugcuguau uggaaacaau acagcauagc 60
aaguuaauau aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugcuuu 119
<210> 185
<211> 12
<212> DNA
<213> Homo sapiens
<400> 185
tagcgggtaa gc 12
<210> 186
<211> 12
<212> DNA
<213> Homo sapiens
<400> 186
tcggtgacat gt 12
<210> 187
<211> 12
<212> DNA
<213> Homo sapiens
<400> 187
actccccgta gg 12
<210> 188
<211> 12
<212> DNA
<213> Homo sapiens
<400> 188
actgcgtgtt aa 12
<210> 189
<211> 12
<212> DNA
<213> Homo sapiens
<400> 189
acgtcgcctg at 12
<210> 190
<211> 12
<212> DNA
<213> Homo sapiens
<400> 190
taggtcgacc ag 12
<210> 191
<211> 12
<212> DNA
<213> Homo sapiens
<400> 191
ggcgttaatg at 12
<210> 192
<211> 12
<212> DNA
<213> Homo sapiens
<400> 192
tgtcgcatgt ta 12
<210> 193
<211> 12
<212> DNA
<213> Homo sapiens
<400> 193
atggaaacgc at 12
<210> 194
<211> 12
<212> DNA
<213> Homo sapiens
<400> 194
gccgaattcc tc 12
<210> 195
<211> 12
<212> DNA
<213> Homo sapiens
<400> 195
gcatggtacg ga 12
<210> 196
<211> 12
<212> DNA
<213> Homo sapiens
<400> 196
cggtactctt ac 12
<210> 197
<211> 12
<212> DNA
<213> Homo sapiens
<400> 197
gcctgtgccg ta 12
<210> 198
<211> 12
<212> DNA
<213> Homo sapiens
<400> 198
tacggtaagt cg 12
<210> 199
<211> 12
<212> DNA
<213> Homo sapiens
<400> 199
cacgaaatta cc 12
<210> 200
<211> 12
<212> DNA
<213> Homo sapiens
<400> 200
aaccaagata cg 12
<210> 201
<211> 12
<212> DNA
<213> Homo sapiens
<400> 201
gagtcgatac gc 12
<210> 202
<211> 12
<212> DNA
<213> Homo sapiens
<400> 202
gtctcacgat cg 12
<210> 203
<211> 12
<212> DNA
<213> Homo sapiens
<400> 203
tcgtcgggtg ca 12
<210> 204
<211> 12
<212> DNA
<213> Homo sapiens
<400> 204
actccgtagt ga 12
<210> 205
<211> 12
<212> DNA
<213> Homo sapiens
<400> 205
caggacgtcc gt 12
<210> 206
<211> 12
<212> DNA
<213> Homo sapiens
<400> 206
tcgtatccct ac 12
<210> 207
<211> 12
<212> DNA
<213> Homo sapiens
<400> 207
tttcaaggcc gg 12
<210> 208
<211> 12
<212> DNA
<213> Homo sapiens
<400> 208
cgccggtgga at 12
<210> 209
<211> 12
<212> DNA
<213> Homo sapiens
<400> 209
gaacccgtcc ta 12
<210> 210
<211> 12
<212> DNA
<213> Homo sapiens
<400> 210
gattcatcag cg 12
<210> 211
<211> 12
<212> DNA
<213> Homo sapiens
<400> 211
acaccggtct tc 12
<210> 212
<211> 12
<212> DNA
<213> Homo sapiens
<400> 212
atcgtgccct aa 12
<210> 213
<211> 12
<212> DNA
<213> Homo sapiens
<400> 213
gcgtcaatgt tc 12
<210> 214
<211> 12
<212> DNA
<213> Homo sapiens
<400> 214
ctccgtatct cg 12
<210> 215
<211> 12
<212> DNA
<213> Homo sapiens
<400> 215
ccgattcctt cg 12
<210> 216
<211> 12
<212> DNA
<213> Homo sapiens
<400> 216
tgcgcctcca gt 12
<210> 217
<211> 12
<212> DNA
<213> Homo sapiens
<400> 217
taacgtcgga gc 12
<210> 218
<211> 12
<212> DNA
<213> Homo sapiens
<400> 218
aaggtcgccc at 12
<210> 219
<211> 12
<212> DNA
<213> Homo sapiens
<400> 219
gtcggggact at 12
<210> 220
<211> 12
<212> DNA
<213> Homo sapiens
<400> 220
ttcgagcgat tt 12
<210> 221
<211> 12
<212> DNA
<213> Homo sapiens
<400> 221
tgagtcgtcg ag 12
<210> 222
<211> 12
<212> DNA
<213> Homo sapiens
<400> 222
tttacgcaga gg 12
<210> 223
<211> 12
<212> DNA
<213> Homo sapiens
<400> 223
aggaagtatc gc 12
<210> 224
<211> 12
<212> DNA
<213> Homo sapiens
<400> 224
actcgatacc at 12
<210> 225
<211> 12
<212> DNA
<213> Homo sapiens
<400> 225
cgctacatag ca 12
<210> 226
<211> 12
<212> DNA
<213> Homo sapiens
<400> 226
ttcataaccg gc 12
<210> 227
<211> 12
<212> DNA
<213> Homo sapiens
<400> 227
ccaaacggtt aa 12
<210> 228
<211> 12
<212> DNA
<213> Homo sapiens
<400> 228
cgattccttc gt 12
<210> 229
<211> 12
<212> DNA
<213> Homo sapiens
<400> 229
cgtcatgaat aa 12
<210> 230
<211> 12
<212> DNA
<213> Homo sapiens
<400> 230
agtggcgatg ac 12
<210> 231
<211> 12
<212> DNA
<213> Homo sapiens
<400> 231
cccctacggc ac 12
<210> 232
<211> 12
<212> DNA
<213> Homo sapiens
<400> 232
gccaacccgc ac 12
<210> 233
<211> 12
<212> DNA
<213> Homo sapiens
<400> 233
tgggacaccg gt 12
<210> 234
<211> 12
<212> DNA
<213> Homo sapiens
<400> 234
ttgactgcgg cg 12
<210> 235
<211> 12
<212> DNA
<213> Homo sapiens
<400> 235
actatgcgta gg 12
<210> 236
<211> 12
<212> DNA
<213> Homo sapiens
<400> 236
tcacccaaag cg 12
<210> 237
<211> 12
<212> DNA
<213> Homo sapiens
<400> 237
gcaggacgtc cg 12
<210> 238
<211> 12
<212> DNA
<213> Homo sapiens
<400> 238
acaccgaaaa cg 12
<210> 239
<211> 12
<212> DNA
<213> Homo sapiens
<400> 239
cggtgtattg ag 12
<210> 240
<211> 12
<212> DNA
<213> Homo sapiens
<400> 240
cacgaggtat gc 12
<210> 241
<211> 12
<212> DNA
<213> Homo sapiens
<400> 241
taaagcgacc cg 12
<210> 242
<211> 12
<212> DNA
<213> Homo sapiens
<400> 242
cttagtcggc ca 12
<210> 243
<211> 12
<212> DNA
<213> Homo sapiens
<400> 243
cgaaaacgtg gc 12
<210> 244
<211> 12
<212> DNA
<213> Homo sapiens
<400> 244
cgtgccctga ac 12
<210> 245
<211> 12
<212> DNA
<213> Homo sapiens
<400> 245
tttaccatcg aa 12
<210> 246
<211> 12
<212> DNA
<213> Homo sapiens
<400> 246
cgtagccatg tt 12
<210> 247
<211> 12
<212> DNA
<213> Homo sapiens
<400> 247
cccaaacggt ta 12
<210> 248
<211> 12
<212> DNA
<213> Homo sapiens
<400> 248
gcgttatcag aa 12
<210> 249
<211> 12
<212> DNA
<213> Homo sapiens
<400> 249
tcgatggtaa ac 12
<210> 250
<211> 12
<212> DNA
<213> Homo sapiens
<400> 250
cgactttttg ca 12
<210> 251
<211> 12
<212> DNA
<213> Homo sapiens
<400> 251
tcgacgactc ac 12
<210> 252
<211> 12
<212> DNA
<213> Homo sapiens
<400> 252
acgcgtcaga ta 12
<210> 253
<211> 12
<212> DNA
<213> Homo sapiens
<400> 253
cgtacggcac ag 12
<210> 254
<211> 12
<212> DNA
<213> Homo sapiens
<400> 254
ctatgccgtg ca 12
<210> 255
<211> 12
<212> DNA
<213> Homo sapiens
<400> 255
cgcgtcagat at 12
<210> 256
<211> 12
<212> DNA
<213> Homo sapiens
<400> 256
aagatcggta gc 12
<210> 257
<211> 12
<212> DNA
<213> Homo sapiens
<400> 257
cttcgcaagg ag 12
<210> 258
<211> 12
<212> DNA
<213> Homo sapiens
<400> 258
gtcgtggact ac 12
<210> 259
<211> 12
<212> DNA
<213> Homo sapiens
<400> 259
ggtcgtcatc aa 12
<210> 260
<211> 12
<212> DNA
<213> Homo sapiens
<400> 260
gttaacagcg tg 12
<210> 261
<211> 12
<212> DNA
<213> Homo sapiens
<400> 261
tagctaaccg tt 12
<210> 262
<211> 12
<212> DNA
<213> Homo sapiens
<400> 262
agtaaaggcg ct 12
<210> 263
<211> 12
<212> DNA
<213> Homo sapiens
<400> 263
ggtaatttcg tg 12
<210> 264
<211> 147
<212> RNA
<213> Artificial Sequence
<220>
<221> source
<223> /note="Description of Artificial Sequence: Synthetic
polynucleotide"
<220>
<221> modified_base
<222> (1)..(20)
<223> a, c, u, g, unknown or other
<400> 264
nnnnnnnnnn nnnnnnnnnn guuuuaguac ucuguaauuu uagguaugag guagacgaaa 60
auuguacuua uaccuaaaau uacagaaucu acuaaaacaa ggcaaaaugc cguguuuauc 120
ucgucaacuu guuggcgaga uuuuuuu 147
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| КОМПОНЕНТЫ СИСТЕМЫ CRISPR-CAS, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796549C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2777988C2 |
| ДОСТАВКА, КОНСТРУИРОВАНИЕ И ОПТИМИЗАЦИЯ СИСТЕМ, СПОСОБОВ И КОМПОЗИЦИЙ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ И ПРИМЕНЕНИЯ В ТЕРАПИИ | 2013 |
|
RU2721275C2 |
| НОВЫЕ ФЕРМЕНТЫ CRISPR И СИСТЕМЫ | 2016 |
|
RU2771826C2 |
| НАПРАВЛЯЕМАЯ РНК РЕГУЛЯЦИЯ ТРАНСКРИПЦИИ | 2014 |
|
RU2756865C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2023 |
|
RU2832308C2 |
| Средство разрезания ДНК на основе Cas9 белка из бактерии Capnocytophaga ochracea | 2021 |
|
RU2778156C1 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2737537C2 |
| СИСТЕМА РЕДАКТИРОВАНИЯ ГЕНОМА CRISPR/CAS9 II ТИПА И ЕЕ ПРИМЕНЕНИЕ | 2022 |
|
RU2794774C1 |
| Средство разрезания двунитевой ДНК с помощью Cas12d белка из Katanobacteria и гибридной РНК, полученной путем слияния направляющей CRISPR РНК и scout РНК | 2020 |
|
RU2771626C1 |
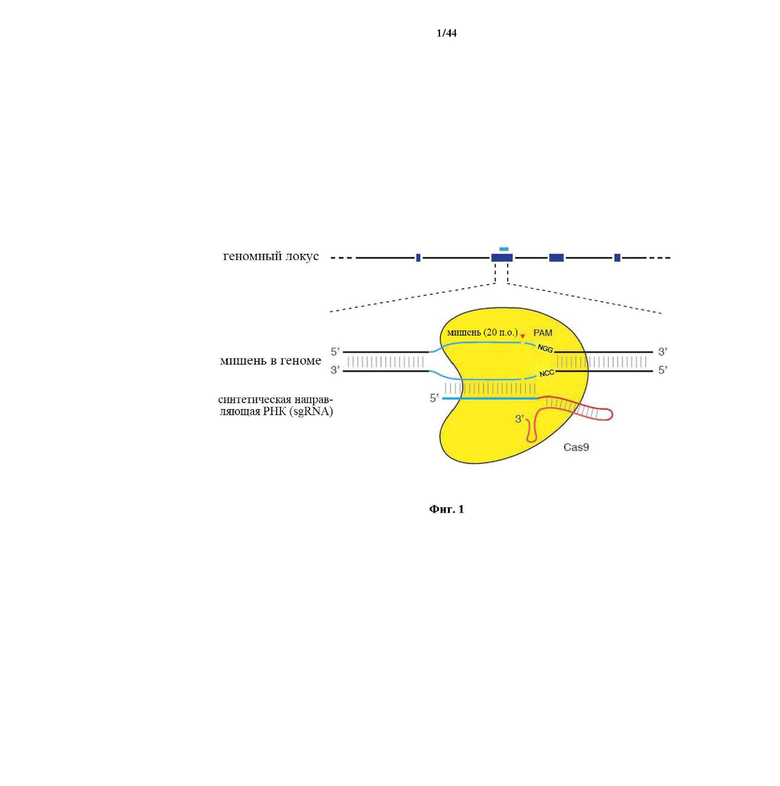

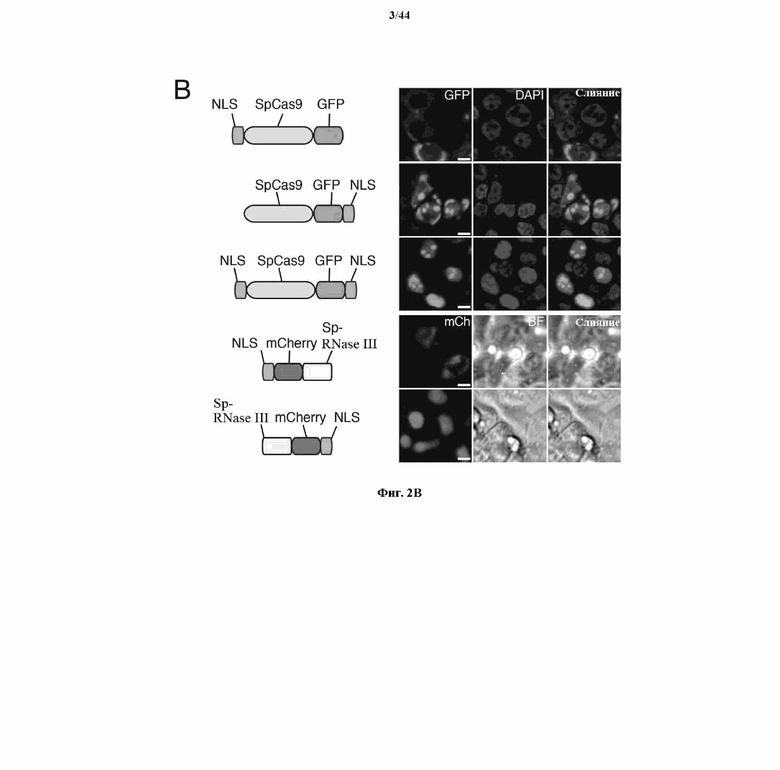

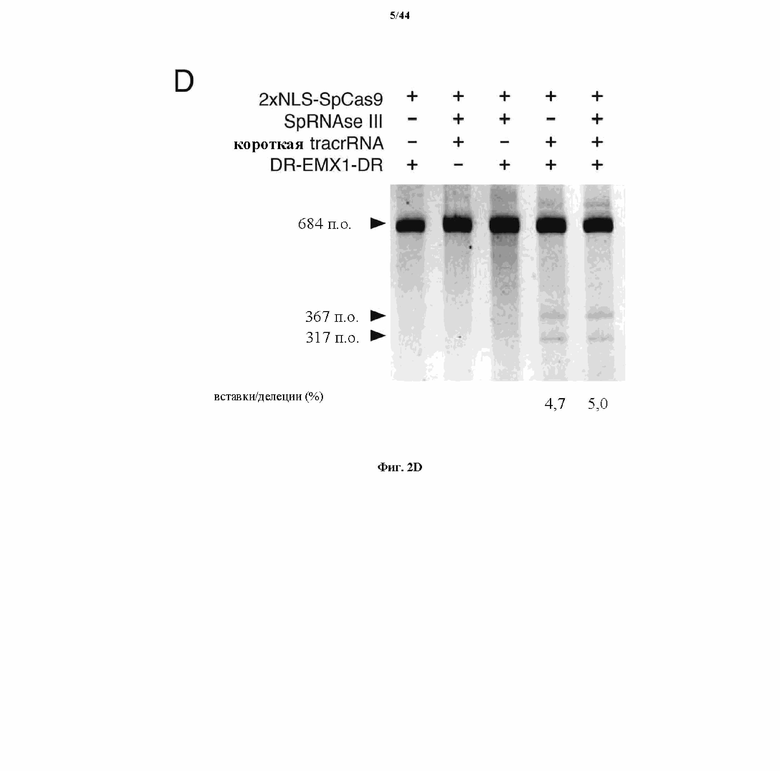

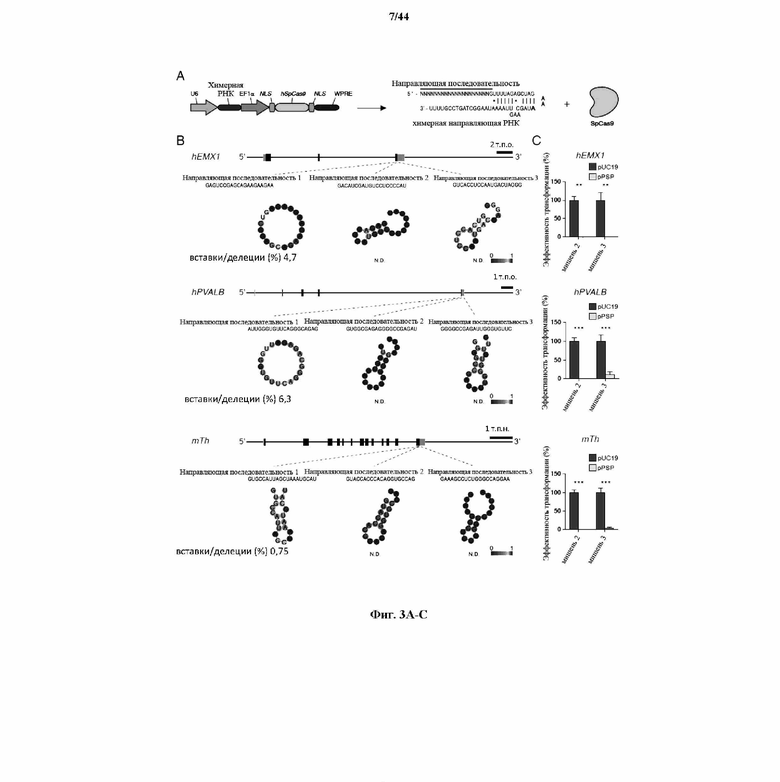
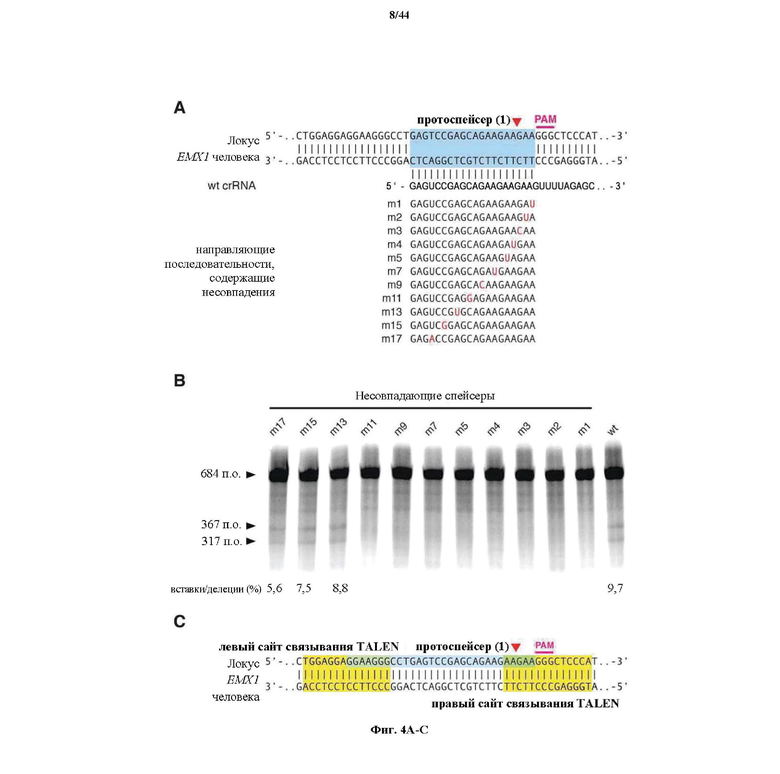
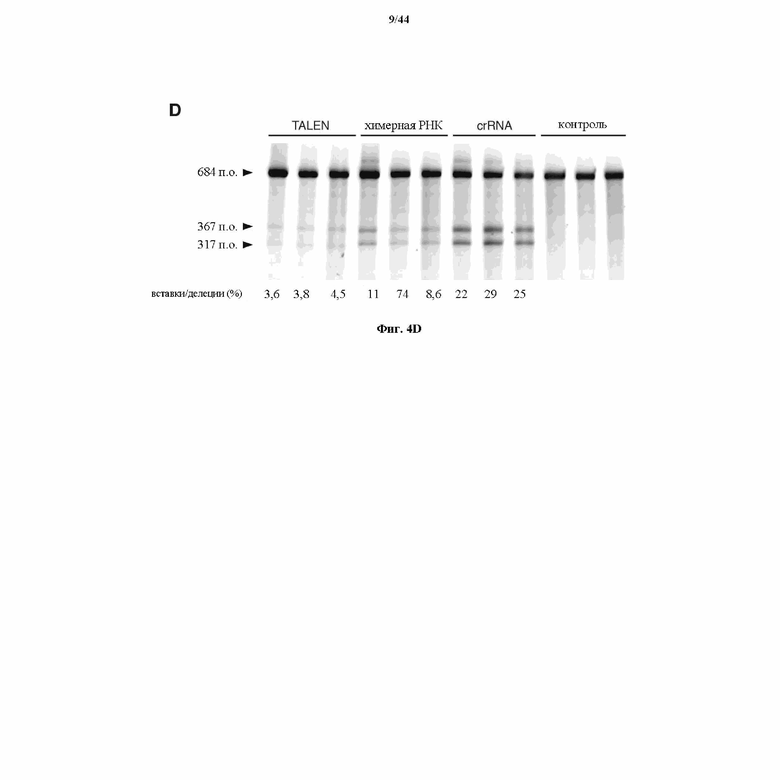

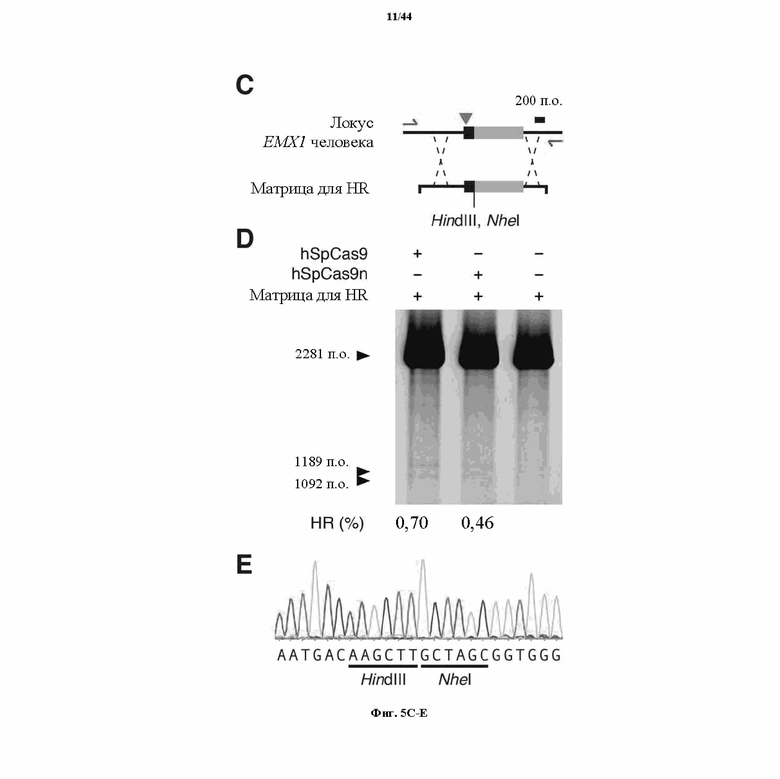
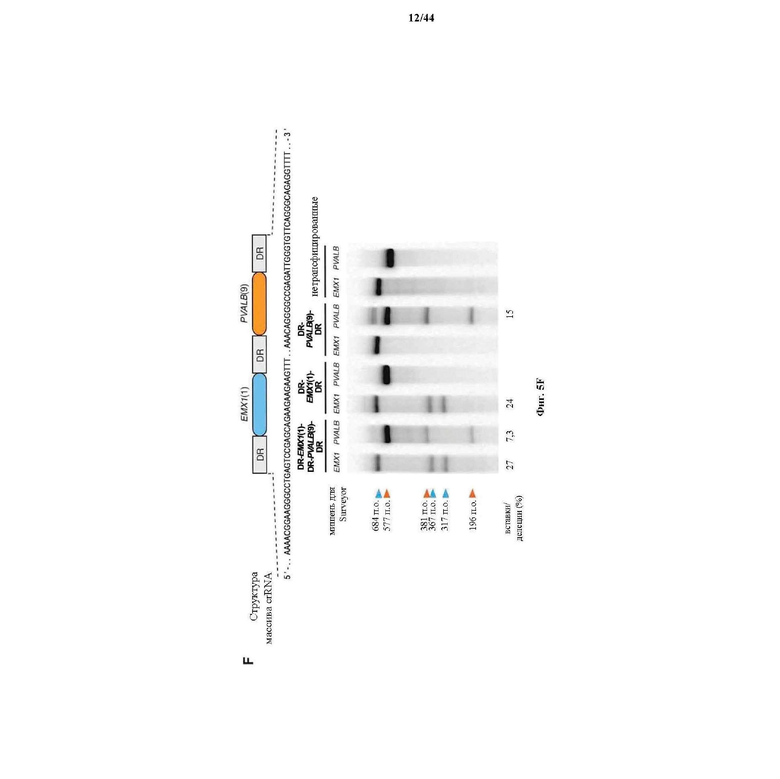

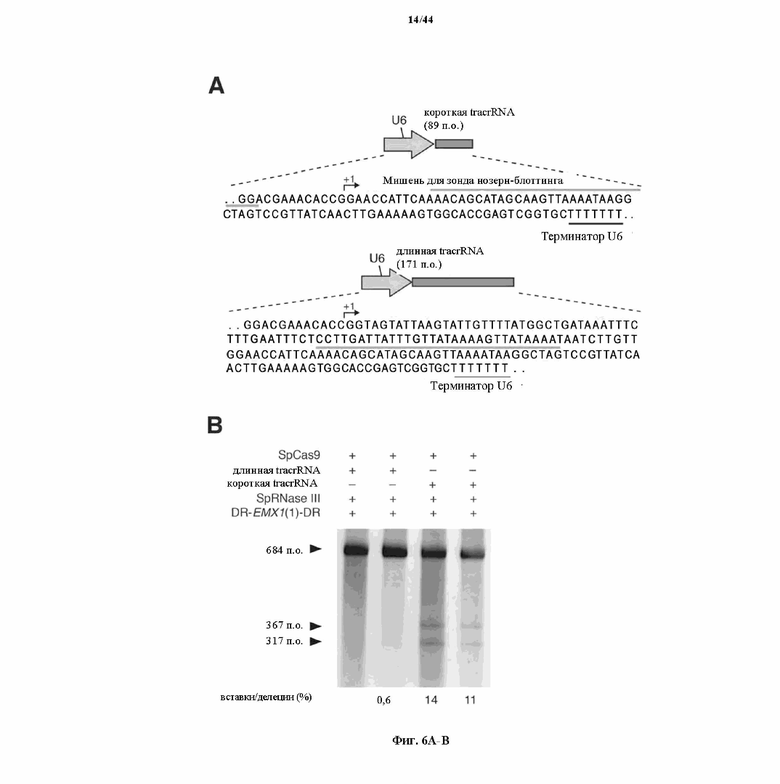
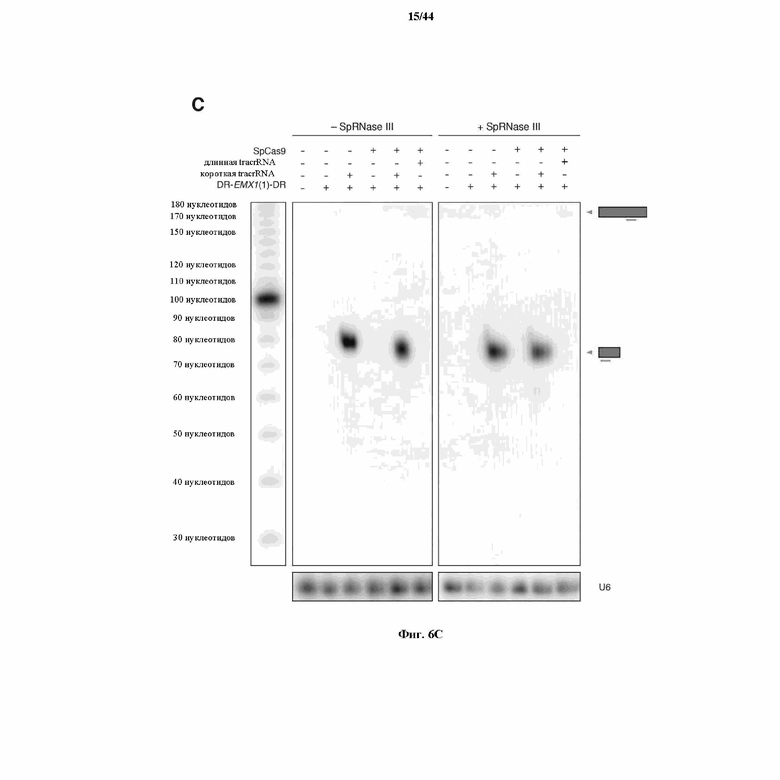
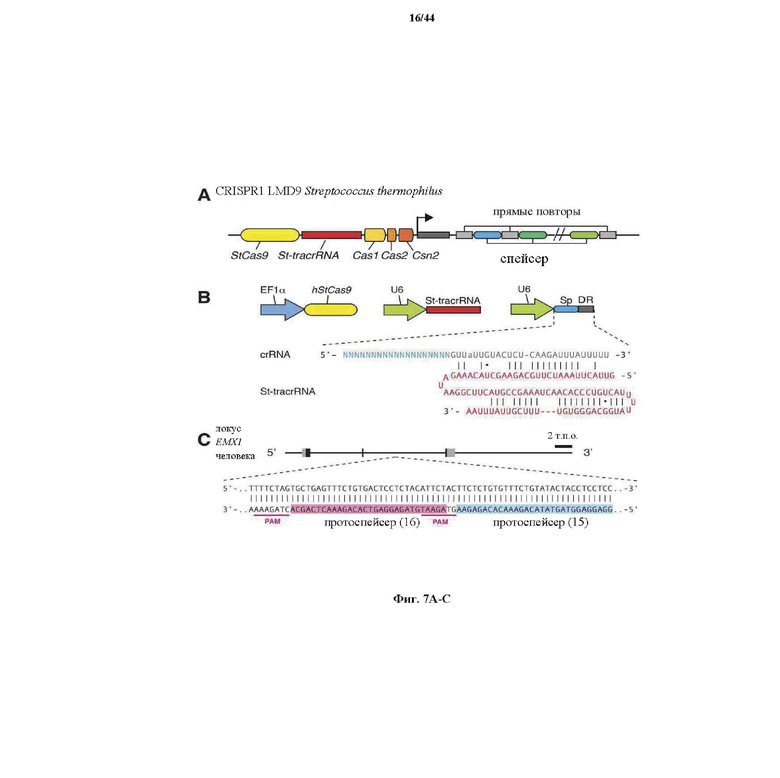
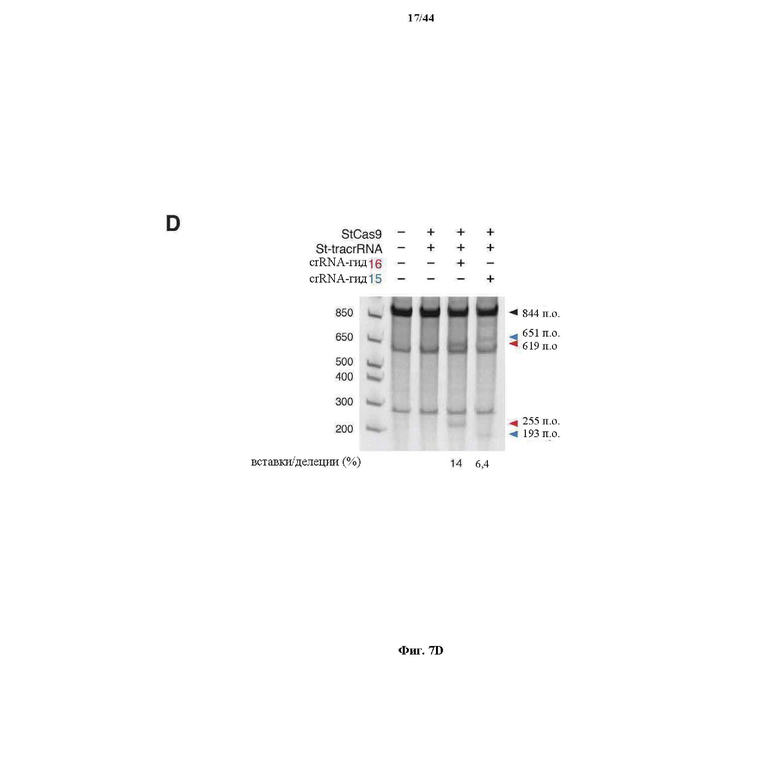

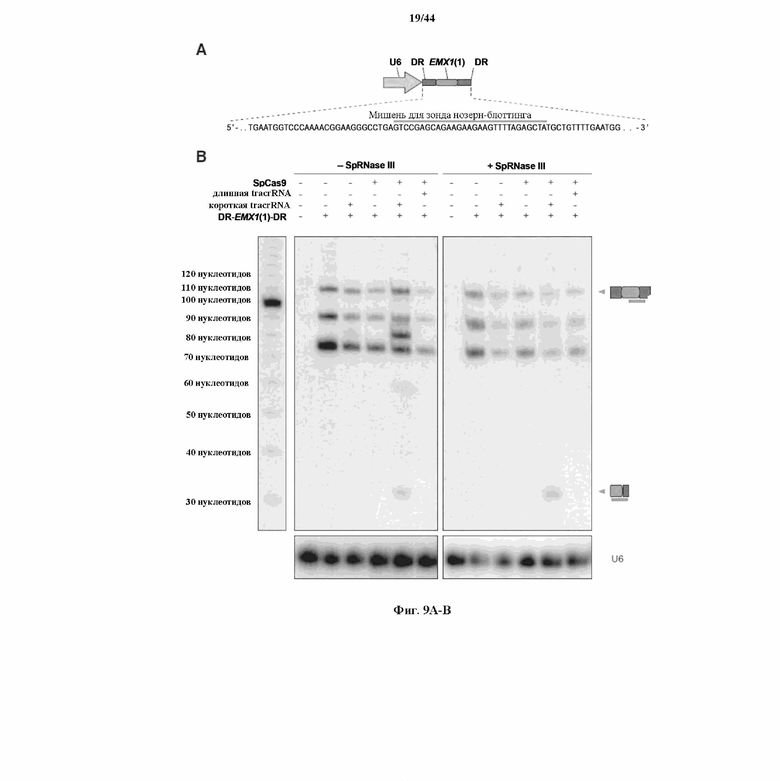
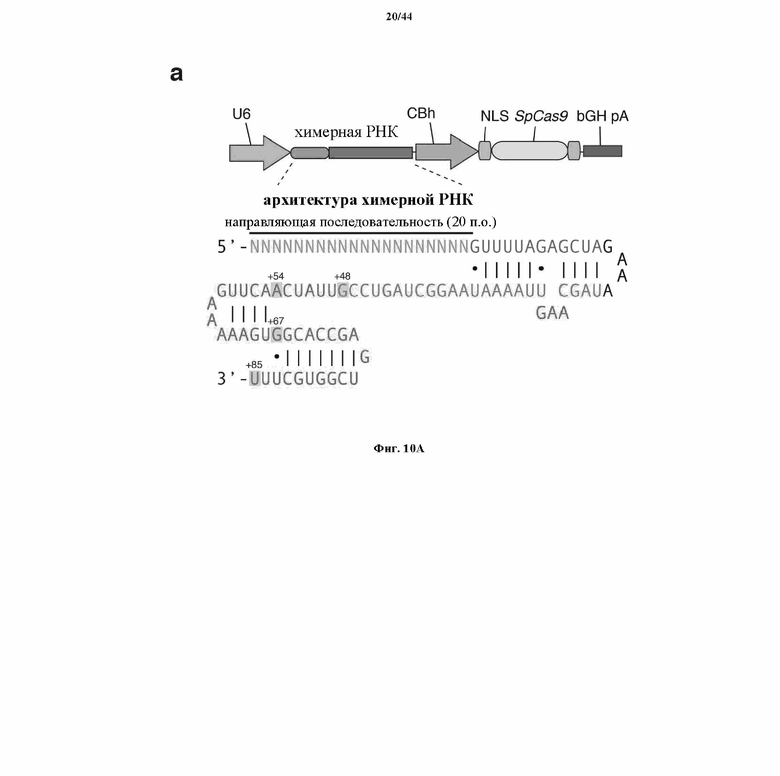

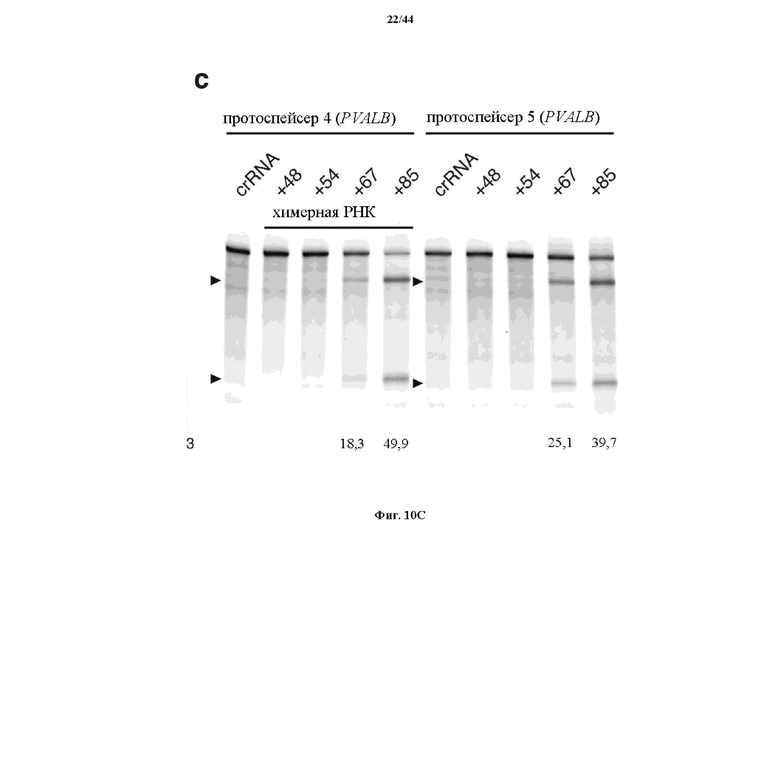

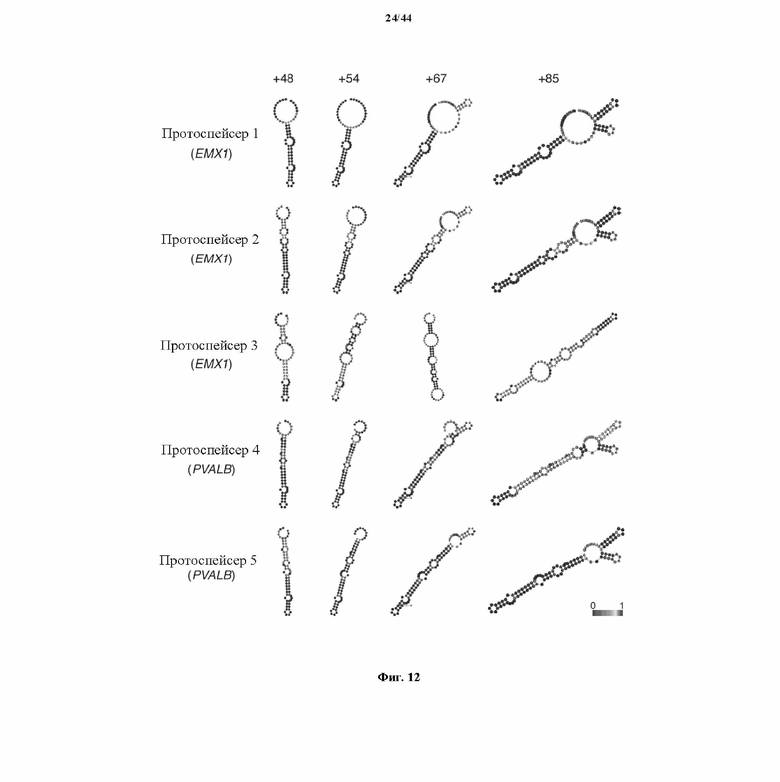




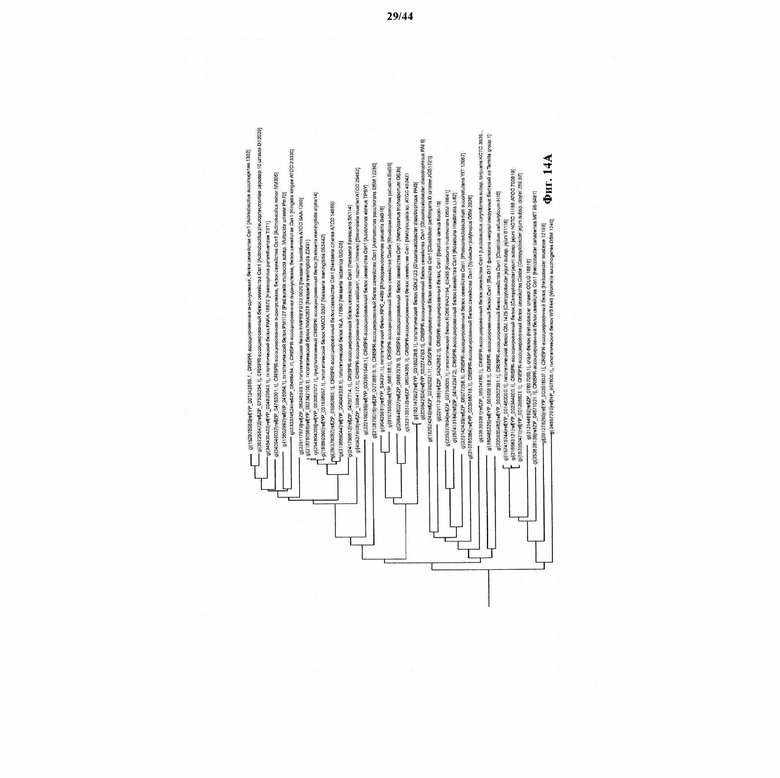

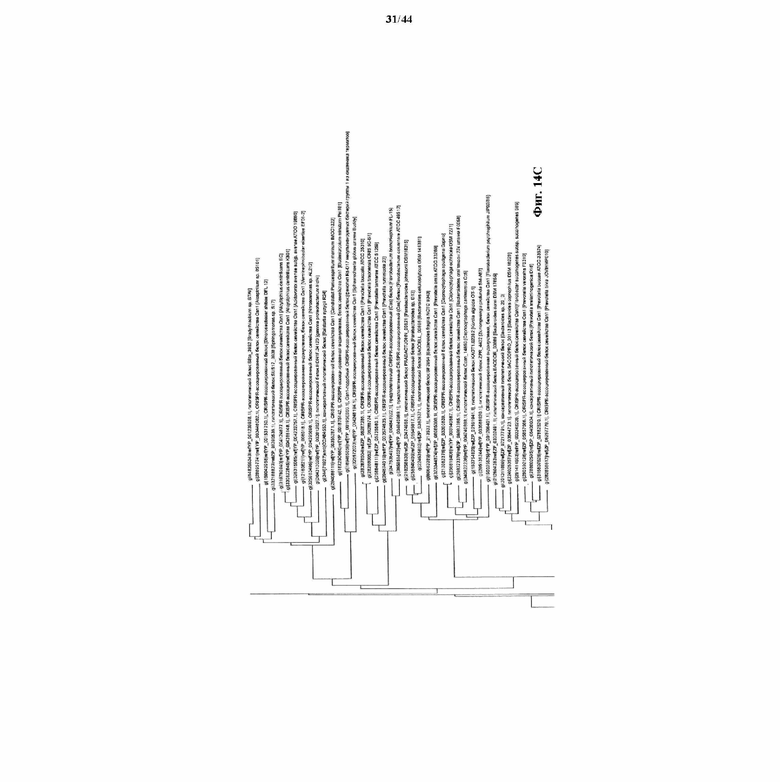
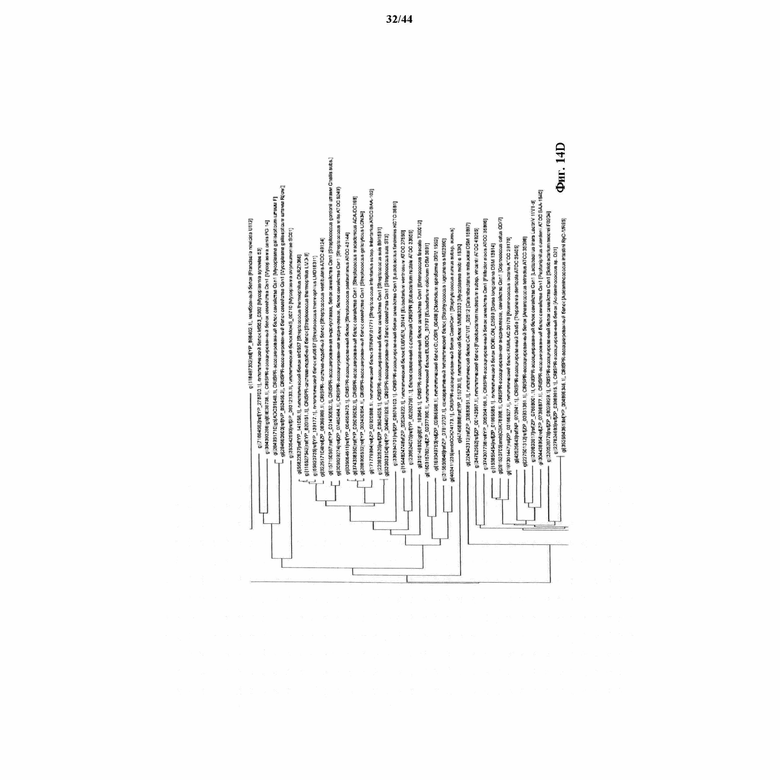


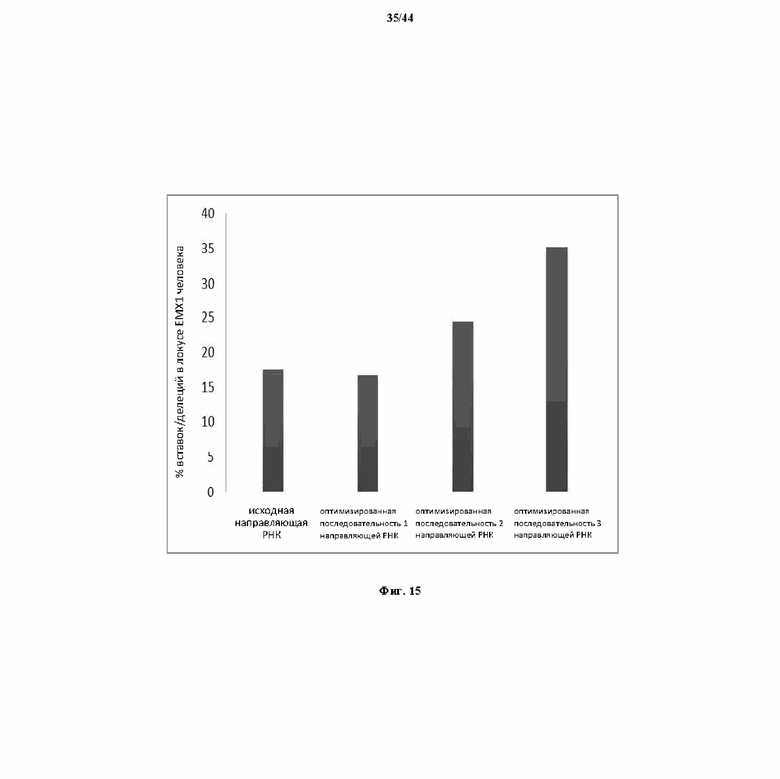

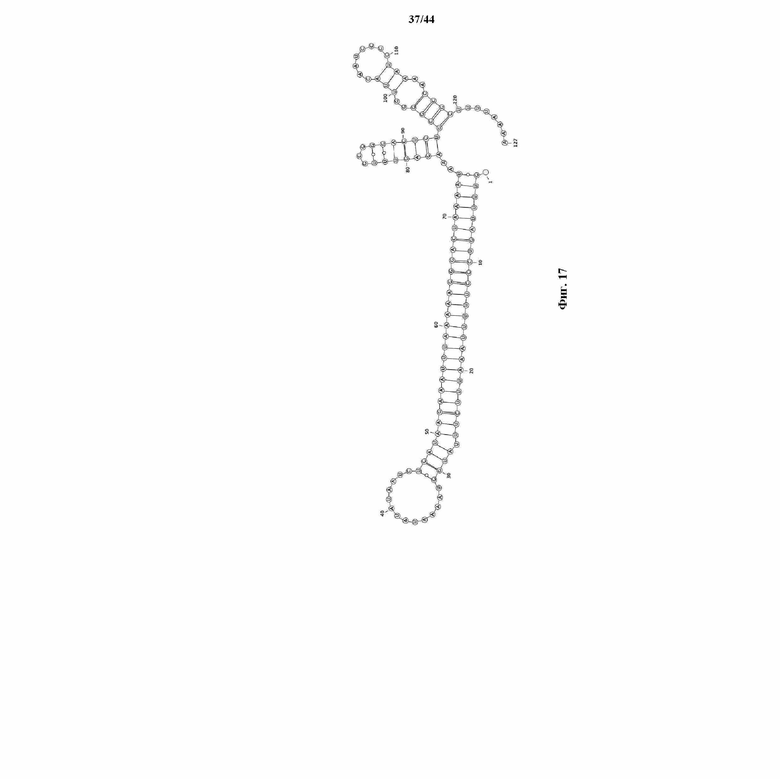
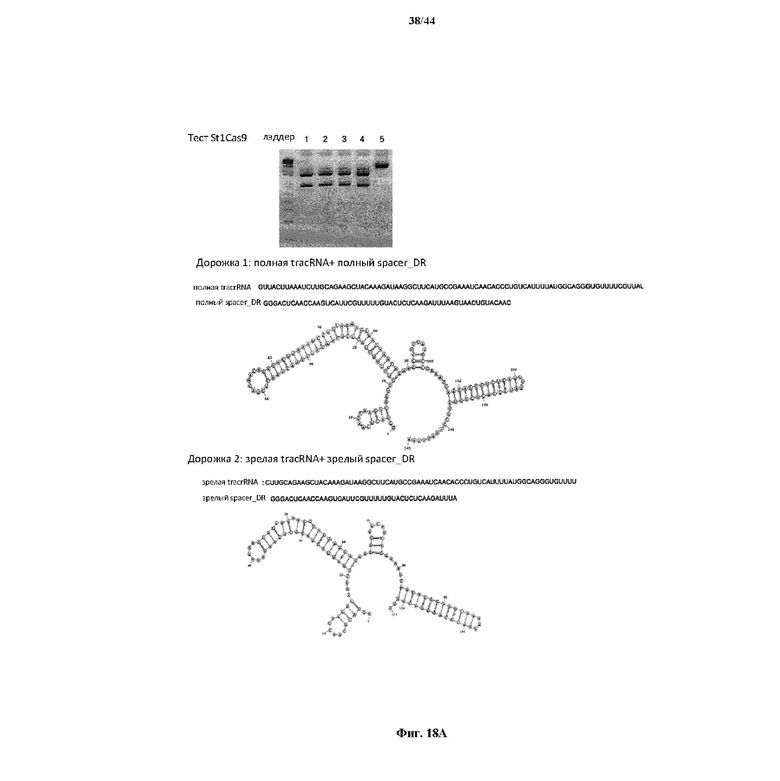


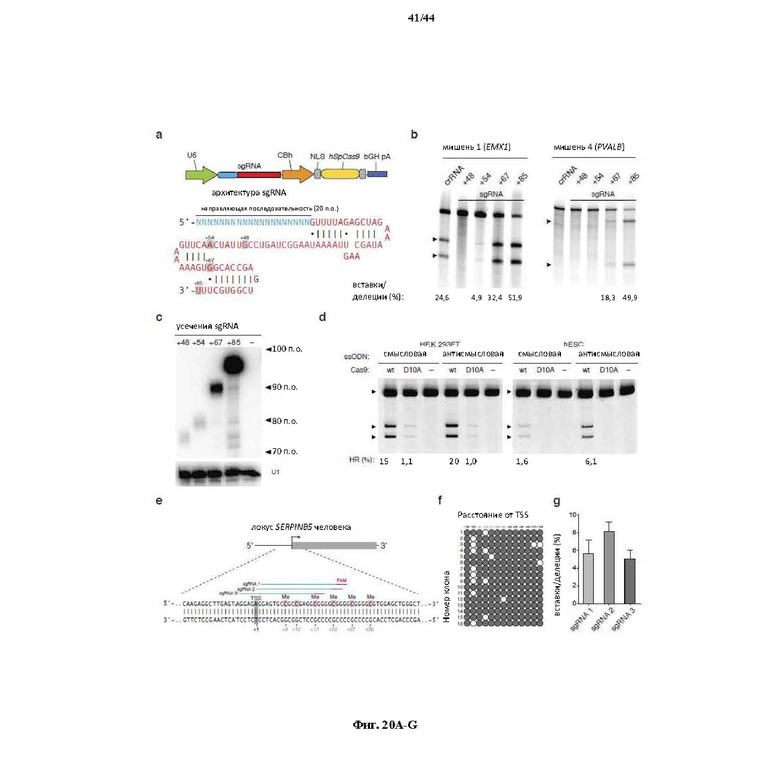
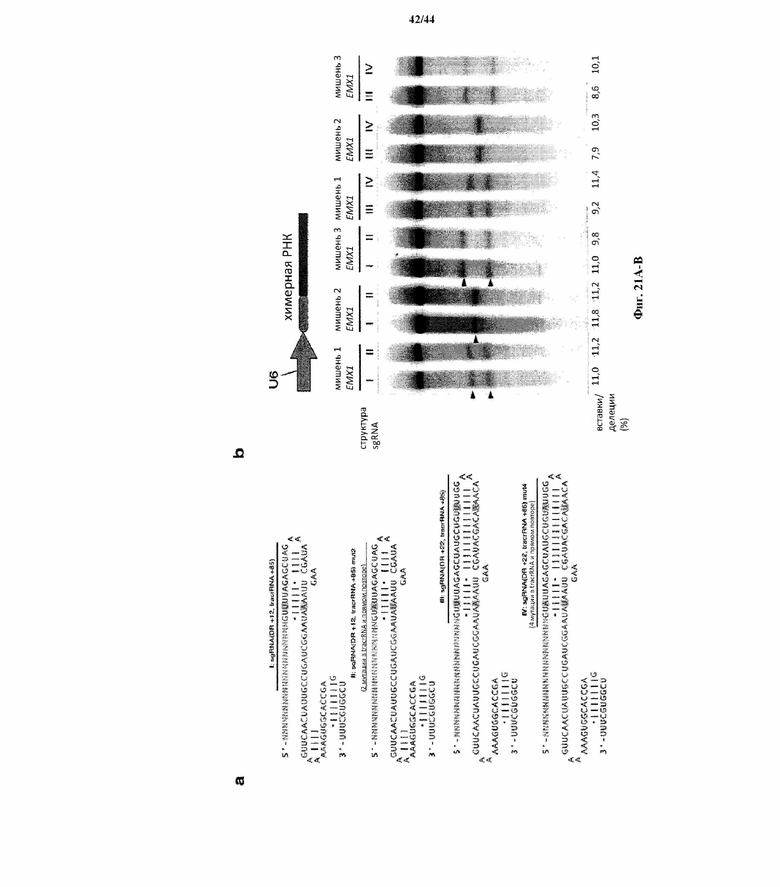

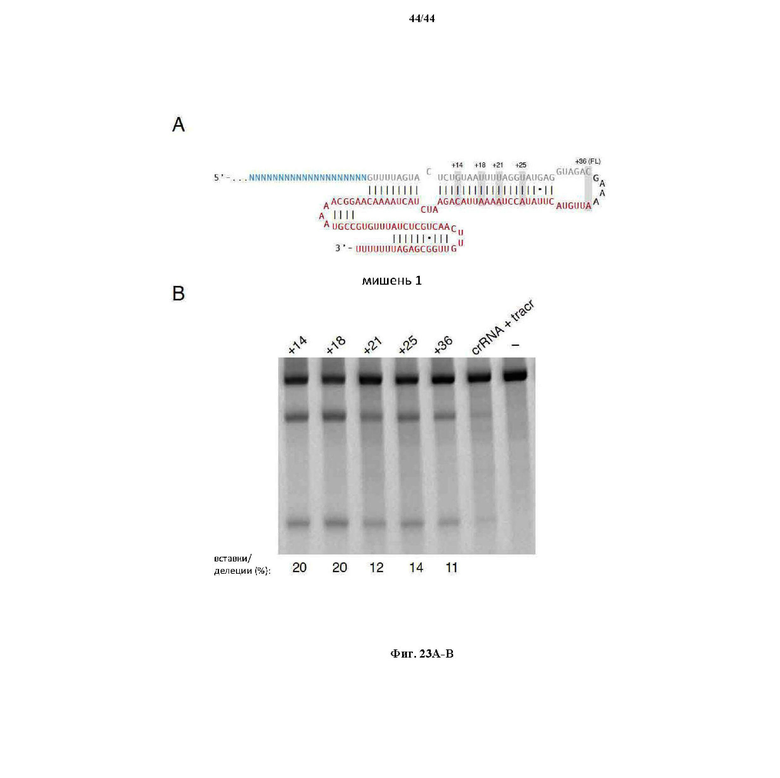
Изобретение относится к области биотехнологии, в частности к вариантам сконструированной системы CRISPR-Cas, содержащей белок Cas9 или одну или более нуклеиновых кислот, кодирующих этот белок Cas9, и первую и вторую РНК системы CRISPR-Cas или одну или более нуклеиновых кислот, кодирующих эти первую и вторую РНК системы CRISPR-Cas. Изобретение эффективно для модификации эукариотической клетки. 2 н. и 48 з.п. ф-лы, 23 ил., 5 табл., 8 пр.
1. Сконструированная система CRISPR-Cas для модификации эукариотической клетки, содержащая:
(a) белок Cas9 или одну или более нуклеиновых кислот, кодирующих этот белок Cas9, и
(b) первую и вторую РНК системы CRISPR-Cas или одну или более нуклеиновых кислот, кодирующих эти первую и вторую РНК системы CRISPR-Cas, где первая РНК системы CRISPR-Cas содержит первую направляющую последовательность, способную гибридизоваться с первой целевой последовательностью, примыкающей к мотиву, смежному с протоспейсером, (PAM), в ядре эукариотической клетки, парную tracr-последовательность, способную гибридизоваться с tracr-последовательностью, и tracr-последовательность, где вторая РНК системы CRISPR-Cas содержит вторую направляющую последовательность, способную гибридизоваться со второй целевой последовательностью, примыкающей к мотиву, смежному с протоспейсером (PAM), в ядре эукариотической клетки, парную tracr-последовательность, способную гибридизоваться с tracr-последовательностью, и tracr-последовательность и где первая целевая последовательность отличается от второй целевой последовательности,
при этом Cas9 слит по меньшей мере с одним сигналом ядерной локализации (NLS), при этом каждая из первой и второй РНК CRISPR-Cas способна образовывать комплекс CRISPR с белком Cas9 и направлять белок Cas9 к целевой последовательности в эукариотической клетке.
2. Система по п. 1, где белок Cas9 слит с по меньшей мере двумя NLS.
3. Система по п.2, в которой белок Cas9 слит с первым NLS на С-конце Cas9 или рядом с ним и со вторым NLS на N-конце Cas9 или рядом с ним.
4. Система по любому из пп.1-3, в которой NLS независимо выбирают из группы, состоящей из PKKKRKV, KRPAATKKAGQAKKKK, PAAKRVKLD, RQRRNELKRSP, NQSSNFGPMKGGNFGGRSSGPYGGGGQYFAKPRNQGGY, RMRIZFKNKGKDTAELRRRRVEVSVELRKAKKDEQILKRRNV, VSRKRPRP, PPKKARED, POPKKKPL, SALIKKKKKMAP, DRLRR, PKQKKRK, RKLKKKIKKL, REKKKFLKRR, KRKGDEVDGVDEVAKKKSKK и RKCLQAGMNLEARKTKK.
5. Система по любому из пп.1-4, в которой Cas9 представляет собой Cas9 S. pyogenes, а PAM включает NGG или где Cas9 представляет собой S. thermophilus Cas9, а PAM включает NNAGAAW.
6. Система по любому из пп.1-5, отличающаяся тем, что Cas9 содержит по меньшей мере одну мутацию в каталитическом домене и представляет собой никазу, не способную расщеплять одну цепь ДНК.
7. Система по п.6, в которой Cas9 содержит по меньшей мере одну мутацию, соответствующую D10A, H840A, N854A или N863A Cas9 S. pyogenes.
8. Система по любому из пп.1-5, отличающаяся тем, что Cas9 содержит по меньшей мере одну мутацию в двух или более каталитических доменах и по существу лишен всей активности расщепления ДНК.
9. Система по п.8, в которой Cas9 содержит мутацию, соответствующую D10A Cas9 S. pyogenes, и дополнительно содержит по меньшей мере одну мутацию, соответствующую H840A, N854A или N863A Cas9 S. pyogenes.
10. Система по любому из пп.1-9, содержащая первую РНК системы CRISPR-Cas и вторую РНК системы CRISPR-Cas, где первая и вторая РНК системы CRISPR-Cas способны направлять белок Cas9 к двум целевым последовательностям, связанным с двумя разными генами, тем самым опосредуя мультиплексную модификацию генома в эукариотической клетке.
11. Система по любому из пп.1-9, содержащая первую РНК системы CRISPR-Cas и вторую РНК системы CRISPR-Cas, где первая и вторая РНК системы CRISPR-Cas способны направлять белок Cas9 к двум целевым последовательностям, фланкирующим геномную область, тем самым опосредуя целенаправленную делецию геномной области в эукариотической клетке.
12. Система по любому из пп.1-11, отличающаяся тем, что каждая из первой и второй РНК системы CRISPR-Cas содержит: crRNA, содержащую направляющую последовательность и парную tracr-последовательность, и tracrRNA, содержащую tracr-последовательность.
13. Система по любому из пп.1-11, в которой каждая из первой и второй РНК системы CRISPR-Cas представляет собой химерную РНК, содержащую в направлении от 5' к 3' направляющую последовательность, парную tracr-последовательность и tracr-последовательность.
14. Система по п.13, в которой tracr-последовательность содержит по меньшей мере 30 нуклеотидов в длину.
15. Система по п.13, в которой tracr-последовательность содержит по меньшей мере 40 нуклеотидов в длину.
16. Система по п.13, в которой tracr-последовательность содержит по меньшей мере 50 нуклеотидов в длину.
17. Система по любому из пп.1-16, отличающаяся тем, что каждая из первой и второй РНК системы CRISPR-Cas содержит один или несколько модифицированных нуклеотидов.
18. Система по любому из пп.1-17, где система содержит первую и вторую РНК системы CRISPR-Cas и белок Cas9.
19. Система по любому из пп.1-17, где система содержит первую и вторую РНК системы CRISPR-Cas и мРНК, кодирующую Cas9, которая кодон-оптимизирована для экспрессии в эукариотической клетке.
20. Система по любому из пп.1-17, где указанные одна или более нуклеиновых кислот, кодирующих белок Cas9, и указанные одна или более нуклеиновых кислот, кодирующих первую и вторую РНК системы CRISPR-Cas, содержатся в одном или нескольких векторах.
21. Система по п.20, в которой указанные одна или более нуклеиновых кислот, кодирующих белок Cas9, и указанные одна или более нуклеиновых кислот, кодирующих первую и вторую РНК системы CRISPR-Cas, содержатся в одном и том же векторе.
22. Система по п.20 или 21, где векторы представляют собой вирусные векторы.
23. Система по п.22, в которой вирусные векторы представляют собой ретровирусные, лентивирусные, аденовирусные, аденоассоциированные или вирусные векторы простого герпеса.
24. Система по любому из пп.1-23, в которой эукариотическая клетка представляет собой клетку млекопитающего или клетку человека.
25. Сконструированная система CRISPR-Cas для модификации эукариотической клетки, включающая:
(a) белок Cas9 или одну или несколько нуклеиновых кислот, кодирующих этот белок Cas9,
(b) РНК системы CRISPR-Cas или одну или несколько нуклеиновых кислот, кодирующих эту РНК системы CRISPR-Cas, где РНК системы CRISPR-Cas содержит направляющую последовательность, способную гибридизоваться с целевой последовательностью, примыкающей к мотиву, смежному с протоспейсером, (PAM) внутри целевой ДНК в ядре эукариотической клетки, парную tracr-последовательность, способную гибридизоваться с tracr-последовательностью, и tracr-последовательность, и
(c) матричный полинуклеотид,
при этом белок Cas9 слит по меньшей мере с одним сигналом ядерной локализации (NLS), при этом РНК системы CRISPR-Cas способна образовывать комплекс CRISPR с белком Cas9 и направлять белок Cas9 к целевой последовательности в эукариотической клетке, и при этом матричный полинуклеотид способен облегчать репарацию разрыва нити, введенного белком Cas9, посредством гомологически направленной репарации.
26. Система по п.25, в которой белок Cas9 слит по меньшей мере с двумя NLS.
27. Система по п.26, в которой белок Cas9 слит с первым NLS на С-конце Cas9 или вблизи него и со вторым NLS на N-конце Cas9 или рядом с ним.
28. Система по любому из пп.25-27, в которой NLS независимо выбирают из группы, состоящей из PKKKRKV, KRPAATKKAGQAKKKK, PAAKRVKLD, RQRRNELKRSP, NQSSNFGPMKGGNFGGRSSGPYGGGGQYFAKPRNQGGY, RMRIZFKNKGKDTAELRRRRVEVSVELRKAKKDEQILKRRNV, VSRKRPRP, PPKKARED, POPKKKPL, SALIKKKKKMAP, DRLRR, PKQKKRK, RKLKKKIKKL, REKKKFLKRR, KRKGDEVDGVDEVAKKKSKK и RKCLQAGMNLEARKTKK.
29. Система по любому из пп.25-28, в которой Cas9 представляет собой Cas9 S. pyogenes, а PAM содержит NGG или где Cas9 представляет собой S. thermophilus Cas9, а PAM включает NNAGAAW.
30. Система по любому из пп.25-29, отличающаяся тем, что Cas9 содержит по меньшей мере одну мутацию в каталитическом домене и представляет собой никазу, не способную расщеплять одну цепь ДНК.
31. Система по п.30, в которой Cas9 содержит по меньшей мере одну мутацию, соответствующую D10A, H840A, N854A или N863A Cas9 S. pyogenes.
32. Система по любому из пп.25-31, отличающаяся тем, что РНК системы CRISPR-Cas содержит: crRNA, содержащую направляющую последовательность и парную tracr-последовательность, и tracrRNA, содержащую tracr-последовательность.
33. Система по любому из пп.25-31, отличающаяся тем, что РНК системы CRISPR-Cas представляет собой химерную РНК, содержащую в направлении от 5' к 3' направляющую последовательность, парную tracr-последовательность и tracr-последовательность.
34. Система по п.33, в которой tracr-последовательность содержит по меньшей мере 30 нуклеотидов в длину.
35. Система по п.33, в которой tracr-последовательность содержит по меньшей мере 40 нуклеотидов в длину.
36. Система по п.33, в которой tracr-последовательность содержит по меньшей мере 50 нуклеотидов в длину.
37. Система по любому из пп.25-36, где РНК системы CRISPR-Cas содержит один или несколько модифицированных нуклеотидов.
38. Система по любому из пп.25-37, в которой матричный полинуклеотид имеет длину не менее 100 нуклеотидов.
39. Система по любому из пп.25-37, в которой матричный полинуклеотид имеет длину не менее 1000 нуклеотидов.
40. Система по любому из пп.25-39, отличающаяся тем, что матричный полинуклеотид перекрывается по меньшей мере с 5 нуклеотидами целевой ДНК.
41. Система по любому из пп.25-39, в которой матричный полинуклеотид перекрывается по меньшей мере с 20 нуклеотидами целевой ДНК.
42. Система по любому из пп.25-40, где репарация приводит к мутации, включающей вставку, делецию или замену одного или нескольких нуклеотидов целевой ДНК.
43. Система по любому из пп.25-40, в которой репарация приводит к интеграции последовательности гена в целевой ДНК.
44. Система по любому из пп.25-43, где система содержит РНК системы CRISPR-Cas и белок Cas9.
45. Система по любому из пп.25-43, где система содержит РНК системы CRISPR-Cas и мРНК, кодирующую Cas9, которая кодон-оптимизирована для экспрессии в эукариотической клетке.
46. Система по любому из пп.25-43, где указанные одна или более нуклеиновых кислот, кодирующих белок Cas9, и указанные одна или более нуклеиновых кислот, кодирующих РНК системы CRISPR-Cas, содержатся в одном или нескольких векторах.
47. Система по п.46, в которой указанные одна или более нуклеиновых кислот, кодирующих белок Cas9, и указанные одна или более нуклеиновых кислот, кодирующих РНК системы CRISPR-Cas, содержатся в одном и том же векторе.
48. Система по п.46 или 47, где векторы представляют собой вирусные векторы.
49. Система по п.48, где вирусные векторы представляют собой ретровирусные, лентивирусные, аденовирусные, аденоассоциированные или вирусные векторы простого герпеса.
50. Система по любому из пп.25-49, где эукариотическая клетка представляет собой клетку млекопитающего или клетку человека.
| MARTIN JINEK et al., A Programmable Dual-RNA-Guided DNA Endonuclease in Adaptive Bacterial Immunity, Science, 2012, 337(6096), 816-821 | |||
| GIEDRIUS GASIUNASA et al., Cas9-crRNA ribonucleoprotein complex mediatesspecific DNA cleavage for adaptive immunityin bacteria, PNAS, 2012, E2579-E2586 | |||
| BLAKE WIEDENHEFT et al., RNA-guided genetic silencing |
Авторы
Даты
2023-05-16—Публикация
2013-12-12—Подача