РОДСТВЕННЫЕ ЗАЯВКИ И ВКЛЮЧЕНИЕ В КАЧЕСТВЕ ССЫЛКИ
[1] По настоящей заявке испрашивается приоритет временной заявке на патент США №62/320231, поданной 8 апреля 2016 года, 62/181675, поданной 18 июня 2015 года, 62/285349, поданной 22 октября 2015 года, 62/296522, поданной 17 февраля 2016 года, и 62/320231, поданной 8 апреля 2016 года.
[2] Все документы, цитируемые или упоминаемые в процитированных в настоящей заявке документах, вместе с любыми инструкциями производителей, описаниями, характеристиками и технологической картой продукта, для любого из продуктов, упомянутых в настоящем описании или в любом документе, включенном в настоящее описание в качестве ссылки, включены в настоящее описание в качестве ссылки и могут быть использованы при осуществлении изобретения. Более конкретно, все упоминаемые документы включены в качестве ссылки в той же степени, как если бы каждый отдельный документ был конкретно и индивидуально указан как включенный в качестве ссылки.
ЗАЯВЛЕНИЕ О ФЕДЕРАЛЬНОМ СПОНСИРОВАНИИ ИССЛЕДОВАНИЯ
[3] Настоящее изобретение было сделано при поддержке правительственных грантов под номерами MH100706, MH110049, DK097768 и GM10407, присужденными Национальным Институтом Здравоохранения. Правительство обладает определенными правами на изобретение.
ОБЛАСТЬ ИЗОБРЕТЕНИЯ
[4] Настоящее изобретение относится к системам, способам и композициям, применяющимся для контроля экспрессии генов, включая нацеливание на последовательности, такое как изменение транскрипции генов или редактирование нуклеиновых кислот, в котором могут использоваться векторные системы, относящиеся к коротким палиндромным повторам, регулярно расположенным кластерами (CRISPR), и их компонентам.
УРОВЕНЬ ТЕХНИКИ, К КОТОРОМУ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
[5] Последние достижения в способах секвенирования генома и анализа получаемых последовательностей существенно увеличили скорость каталогизации и картирования генетических факторов, ассоциированных с широким спектром биологических функций и заболеваний. Технологии направленного внесения изменений в геном необходимы для систематического поиска генетических вариаций, являющихся причинами заболеваний, а также для развития синтетической биологии, применений в биотехнологии и медицине. Несмотря на существование различных техник редактирования генома, таких как сконструированные цинковые пальцы, подобные активатору транскрипции эффекторы (TALE), хоминг-мегануклеазы, остается потребность в новых технологиях инженерии генома и транскриптома, в которых используются новейшие стратегии и молекулярные механизмы, и которые являются доступными, легко устанавливаемыми, масштабируемыми и способными к нацеливанию на множественно положений в пределах эукариотического генома и транскриптома. Это может обеспечить основной ресурс для новых подходов в генной инженерии и биотехнологии.
[6] Системы адаптивного иммунитета бактерий и архей CRISPR-Cas демонстрируют огромное разнообразие белкового состава и архитектуры геномных локусов. Локусы системы CRISPR-Cas включают более 50 семейств генов, и не существует строго универсальных генов, что указывает на быструю эволюцию и чрезвычайное разнообразие архитектуры локусов. На данный момент с использованием комплексного подхода примерно 395 профилей 93 Cas-белков были исчерпывающе идентифицированы как гены cas. Для классификации систем CRISPR-Cas используют профили генов и характерные особенности архитектуры локусов. В последней предложенной классификации системы CRISPR-Cas в самом общем виде разделены на два класса: системы 1 класса состоят из многосубъединичных эффекторных комплексов, а в системах 2 класса эффекторный модуль состоит из одного белка, например, белка Cas9 (фиг. 1A и 1B). Новые эффекторные белки, ассоциированные с системами CRISPR-Cas 2 класса, могут служить мощными инструментами для геномной инженерии и прогнозирование предполагаемых новых эффекторных белков и их конструирование и оптимизация представляются важными.
[7] Цитирование или указание на какой-либо документ в настоящей заявке не является признанием того, что такой документ доступен в качестве уровня техники для настоящего изобретения.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
[8] CRISPR-Cas является системой адаптивного иммунитета, которая защищает микроорганизмы от чужеродных генетических элементов путем интерференции ДНК или РНК-ДНК. Авторы изобретения изучили принадлежащий ко 2 классу типа IV однокомпонентный эффектор C2c2 CRISPR-Cas и охарактеризовали его как РНК-направленную РНК-азу. Авторы изобретения продемонстрировали, что C2c2 (к примеру, выделенный из Leptotrichia shahii) вызывает сильную интерференцию в ответ на заражение РНК-содержащим фагом. Путем биохимического анализа in vitro и анализов in vivo, авторы изобретения показали, что C2c2 может быть запрограммирован для расщепления оцРНК-мишеней, несущих протоспейсеры, фланкированные 3'-H (не G) последовательностями. Расщепление опосредуется каталитическими остатками в двух консервативных доменах HEPN C2c2, мутации в которых приводят к каталитически неактивному РНК-связывающему белку. C2c2 направляется единичной cr-РНК и может быть перепрограммирован для удаления конкретных мРНК in vivo. Авторы изобретения показали, что LshC2c2 может быть нацелен на конкретный представляющий интерес участок и может проявлять неспецифичную РНК-азную активность после стимуляции его РНК-мишенью. Эти результаты расширяют понимание систем CRISPR-Cas и демонстрируют возможность использования C2c2 для разработки широкого арсенала инструментов нацеливания на РНК.
[9] Существует острая необходимость разработки альтернативных и надежных систем и техник для нацеливания на нуклеиновые кислоты и полинуклеотиды (например, ДНК или РНК, а также любые их гибриды и производные) с широким спектром практических применений. Настоящее изобретение направлено на решение этой задачи и имеет ряд связанных с этим преимуществ. Обогащение существующего репертуара технологий нацеливания на представляющую интерес последовательность ДНК или РНК (таргетинга) в геноме и эпигеноме новыми системами, представленными в настоящем описании, может привести к преобразованиям не только в изучении, но и в изменении или редактировании определенных участков-мишеней путем прямого обнаружения, анализа и модификации. Чтобы эффективно использовать рассматриваемые в данной заявке системы геномного или эпигеномного нацеливания на ДНК или РНК без неблагоприятных последствий, важно понимать основные принципы инженерии и оптимизации этих инструментов нацеливния на ДНК или РНК.
[10] Эффекторный белок C2c2, относящийся к типу VI класса 2, является РНК-направленной РНК-азой, которая может быть эффективно запрограммирована на деградацию оцРНК. C2c2 производит отщепление РНК посредством консервативных основных остатков внутри двух его HEPN-доменов, в отличие от каталитических механизмов, известных для других РНК-аз, обнаруженных в системах CRISPR-Cas. Мутации в HEPN-домене, такие как замена (например, на аланин) в любом из четырех предсказанных каталитических остатков HEPN-домена превращает C2c2 в неактивный программируемый РНК-связывающий белок (dC2c2, аналогичный dCas9).
[11] Способность С2с2 связываться с определенными последовательностями может быть использована в нескольких аспектах согласно изобретению: (i) для доставки эффекторных модулей к определенным транскриптам с целью модулирования их функции или трансляции, что может быть использовано для крупномасштабного скрининга, конструирования синтетических регуляторных систем и других целей, (ii) для флуоресцентного мечения определенных РНК для визуализации их перемещений и/или локализации, (iii) для изменения локализации РНК посредством доменов с аффинностью к конкретным внутриклеточным компартментам, и (iv) для захвата определенных транскриптов (посредством их прямого связывания с C2c2 или с использованием C2c2 для локализации активности биотин-лигазы к определенным транскриптам) для увеличения содержания проксимальных молекулярных партнеров, включая РНК и белки.
[12] Активный C2c2 имеет большие перспективы практического применения. Одним из аспектов настоящего изобретения является нацеливание на конкретный транскрипт для разрушения, как в случае с RFP, описанного в настоящем описании. Кроме того, после стимуляции собственной мишенью C2c2 может расщеплять другие (некомплементарные) молекулы РНК in vitro и ингибировать рост клеток in vivo. В биологическом смысле, эта неспецифическая РНК-азная активность может находить отражение в защитном механизме систем CRISPR-Cas типа VI на основе запрограммированной клеточной смерти/покое PCD/D. Соответственно, в одном аспекте изобретения ее можно использовать для запуска PCD или покоя в конкретных клетках, например, злокачественных клетках, экспрессирующих определенный транскрипт, нейронах определенного класса, клетках, зараженных определенным патогеном, или других аберрантных клетках, присутствие которых нежелательно.
[13] Настоящее изобретение относится к способу модификации последовательностей нуклеиновых кислот, ассоциированных с локусом-мишенью или находящихся непосредственно в нем, причем способ включает доставку в указанный локус не встречающейся в природе или сконструированной композиции, содержащей эффекторный белок локусов системы CRISPR-Cas типа VI и один или более компонентов-нуклеиновых кислот, где эффекторный белок формирует комплекс с одним или более компонентами-нуклеиновыми кислотами и при связывании указанного комплекса с представляющим интерес локусом эффекторный белок индуцирует модификацию последовательностей, ассоциированных с представляющим интерес локусом или находящихся непосредственно в нем. В предпочтительном варианте осуществления модификация представляет собой внесение разрыва цепи. В предпочтительном варианте осуществления последовательности в локусе-мишени, ассоциированные с ним или находящиеся непосредственно в нем, включают РНК или ДНК, и эффекторный белок кодируется локусами CRISPR-Cas типа VI.
[14] Будет понятно, что термины "фермент Cas", "фермент CRISPR", "белок CRISPR", "белок Cas" и "CRISPR Cas" используются обычно взаимозаменяемо и в каждом случае, когда они упоминаются в настоящем описании, обозначают новые эффекторные белки CRISPR, описанные далее в настоящем описании, если иное не следует из контекста, в частности при конкретном упоминании Cas9. Эффекторные белки CRISPR, описанные в настоящем описании, предпочтительно являются эффекторными белками C2c2.
[15] Изобретение относится к способу модификации последовательностей, ассоциированных с или находящихся в представляющем интерес локусе-мишени, причем способ включает доставку в указанные последовательности, ассоциированные с или находящиеся в локусе не встречающейся в природе или сконструированной способами инженерии композиции, содержащей эффекторный белок локусов C2c2 и один или более компонентов, являющихся нуклеиновыми кислотами, где эффекторный белок C2c2 образует комплекс с одним или более компонентами, являющимися нуклеиновыми кислотами, и при связывании указанного комплекса с представляющим интерес локусом эффекторный белок индуцирует модификацию последовательностей, ассоциированных с или находящихся в представляющем интерес локусе-мишени. В предпочтительном варианте осуществления модификация представляет собой внесение разрыва цепи. В предпочтительном варианте осуществления эффекторный белок C2c2 образует комплекс с одним компонентом, являющимся - нуклеиновой кислотой, преимущественно сконструированным способами инженерии или не встречающимся в природе компонентом, являющимся нуклеиновой кислотой. Индукция модификации последовательностей, ассоциированных с или находящихся в представляющем интерес локусе-мишени, может направляться комплексом эффекторный белок C2c2-нуклеиновая кислота. В предпочтительном варианте осуществления изобретения указанным компонентом, являющимся нуклеиновой кислотой, является РНК CRISPR (cr-RNA). В предпочтительном варианте осуществления компонентом, являющимся нуклеиновой кислотой, является зрелая cr-РНК или направляющая РНК, где зрелая cr-РНК или направляющая РНК содержит спейсерную последовательность (или направляющую последовательность) и содержащую прямые повторы последовательность либо ее производные. В предпочтительном варианте осуществления спейсерная последовательность или ее производное включает последовательность-затравку, причем последовательность-затравка имеет критическое значение для распознавания и/или гибридизации с последовательностью локуса-мишени. В предпочтительном варианте осуществления изобретения последовательности, ассоциированные с или находящиеся в целевом представляющем интерес локусе, содержат линейную или суперспиральную ДНК.
[16] Аспекты изобретения связаны с образованными эффекторным белком C2c2 комплексами, включающими один или более компонентов, являющихся не встречающимися в природе, или сконструированными способами инженерии, или модифицированными, или оптимизированными нуклеиновыми кислотами. В предпочтительном варианте осуществления изобретения компонент, являющийся нуклеиновой кислотой, комплекса может содержать направляющую последовательность, связанную с содержащей прямые повторы последовательностью, где содержащая прямые повторы последовательность содержит одну или более шпилечных структур или оптимизированные вторичные структуры. В определенных вариантах осуществления изобретения прямые повторы имеют длину минимум 16 п.н., такую как не менее 28 п.н., а также одну шпилечную структуру. В следующих вариантах осуществления изобретения прямые повторы могут иметь длину более 16 п.н., предпочтительно 17 п.н., такую как не менее 28 п.н., а также одну или более шпилечных структур или оптимизированных вторичных структур. В конкретных вариантах осуществления изобретения прямой повтор может быть модифицирован таким образом, чтобы он содержал один или более связывающих белки РНК-аптамеров. В предпочтительном варианте осуществления изобретения один или более аптамеров могут быть включены в качестве части оптимизированной вторичной структуры. Такие аптамеры могут быть способны к связыванию белка оболочки бактериофага. Белок оболочки бактериофага может быть выбран из группы, включающей Qβ, F2, GA, fr, JP501, MS2, M12, R17, BZ13, JP34, JP500, KU1, M11, MX1, TW18, VK, SP, FI, ID2, NL95, TW19, AP205, ϕCb5, ϕCb8r, ϕCb12r, ϕCb23r, 7s и PRR1. В предпочтительном варианте осуществления изобретения используется белок оболочки бактериофага MS2. Изобретение также относится к компонентам вышеназванных комплексов, являющимся нуклеиновыми кислотами длиной 30 или более, 40 или более, а также 50 или более п.н.
[17] Изобретение относится к способам редактирования генома и изменения транскриптома, причем способ включает два или более этапа нацеливания на эффекторный белок C2c2 и расщепления. В определенных вариантах осуществления изобретения первый этап включает расщепление эффекторным белком C2c2 последовательностей, ассоциированных с локусом-мишенью, находящимся на большом расстоянии от последовательности-затравки, в то время как второй этап включает расщепление эффекторным белком С2с2 последовательностей в локусе-мишени. В определенных таких вариантах осуществления изобретения первый этап нацеливания эффекторного белка C2c2 приводит к образованию разрыва цепи, в то время как второй этап нацеливания эффекторного белка C2c2 приводит к второму разрыву цепи. В одном из вариантов осуществления изобретения один или более этапов нацеливания эффекторного белка C2c2 приводит к образованию разрыва с липкими концами, который может быть репарирован.
[18] Изобретением также относится к способу модификации представляющего интерес локуса-мишени, причем способ включает доставку в указанный локус не встречающейся в природе или сконструированной способами инженерии композиции, содержащей эффекторный белок локусов С2с2 и один или более компонентов, являющихся нуклеиновыми кислотами, где эффекторный белок С2с2 образует комплекс с одним или более компонентами, являющимися нуклеиновой кислотой, и при связывании указанного комплекса с представляющим интерес локусом эффекторный белок индуцирует модификацию представляющего интерес локуса-мишени. В предпочитаемом варианте осуществления изобретения такой модификацией является разрыв цепи.
[19] В таких способах представляющий интерес локус-мишень может являться частью молекулы РНК. Помимо этого, представляющий интерес локус-мишень может являться частью молекулы ДНК, в определенных вариантах осуществления изобретения - частью транскрибированной молекулы ДНК. В таких способах представляющий интерес локус-мишень может быть частью молекулы нуклеиновой кислоты in vitro.
[20] В таких способах представляющий интерес локус-мишень может быть частью молекулы нуклеиновой кислоты в клетке. Такая клетка может быть как прокариотической, так и эукариотической. Такая клетка может быть клеткой млекопитающего. Такая клетка млекопитающего может быть клеткой отличного от человека примата, клеткой коровы, свиньи, грызуна или мыши. Такая клетка может быть клеткой домашней птицы, рыбы или креветки. Такая клетка может быть растительной клеткой, полученной из таких растений как маниок, кукуруза, сорго, пшеница или фиг. Такая клетка растения может также быть клеткой водоросли, дерева или овоща. Модификации, внесенные в такую клетку в соответствии с настоящим изобретением, могут быть выполнены таким образом, что такая клетка и ее потомки будут изменены с целью улучшить производство биологических продуктов, таких как антитела, крахмал, спирт или другой желаемый продукт клетки. Такие внесенные в клетку модификации в соответствии с настоящим изобретением могут быть способны приводить к тому, что клетка или ее дочерние клетки будут иметь свойство производить измененный продуцируемый биологический продукт.
[21] Клетка млекопитающего может быть клеткой отличного от человека млекопитающего, например, клеткой примата, коровы, овцы, свиньи, собаки, грызуна, представителя семейства Leporidae, клеткой такого млекопитающего как обезьяна, клеткой коровы, овцы, свиньи, собаки, кролика, крысы или мыши. Клетка может быть эукариотической клеткой не являющегося млекопитающим животного, такого как домашняя птица (например, курицы), позвоночной рыбы (например, лосося) или беспозвоночного (например, устрицы, двустворчатого моллюска, лобстера, креветки). Такая клетка может быть также клеткой растения. Такая растительная клетка может быть получена из однодольного растения или двудольного растения, а также сельскохозяйственного растения или злака, такого как маниок, кукуруза, сорго, соя, пшеница, овес или фиг. Такая растительная клетка может быть также быть клеткой водоросли, дерева или растения-продуцента, фрукта или овоща (например, дерева такого как цитрусовые деревья, например, апельсина, грейпфрута или лимона; персика или нектарина; яблони и груши; орехоносного дерева, такого как миндаль, грецкий орех или фисташка; пасленового растения; растения рода Brassica; растения рода Lactuca; растения рода Spinacia; растения рода Capsicum; хлопка, табака, спаржи, моркови, капусты, брокколи, цветной капусты, томата, баклажана, перца, латука, шпината, клубники, черники, малины, ежевики, винограда, кофе, какао и т.д.)
[22] Изобретение относится к способу модификации представляющего интерес локуса-мишени, причем способ включает доставку в указанный локус не встречающейся в природе или сконструированной способами инженерии композиции, содержащей эффекторный белок локусов CRISPR-Cas типа IV и один или более компонентов, являющихся нуклеиновыми кислотами, где эффекторный белок образует комплекс с одним или более компонентами, являющимися нуклеиновыми кислотами, причем после связывания указанного комплекса с представляющим интерес локусом эффекторный белок индуцирует модификацию представляющего интерес локуса-мишени. В предпочитаемом варианте осуществления изобретения такой модификацией является разрыв цепи.
[23] В таких способах представляющий интерес локус-мишень может являться частью молекулы ДНК или молекулы РНК. В предпочитаемом варианте осуществления изобретения представляющий интерес локус-мишень представляет собой РНК.
[24] Настоящее изобретение также относится к способу модификации представляющего интерес локуса-мишени, причем способ включает доставку в указанный локус не встречающейся в природе или сконструированной способами инженерии композиции, содержащей эффекторный белок локуса C2c2 и один или более компонентов, являющихся нуклеиновыми кислотами, где эффекторный белок C2c2 формирует комплекс с одним или более компонентами, являющимися нуклеиновыми кислотами и при связывании указанного комплекса с представляющим интерес локусом эффекторный белок индуцирует модификацию представляющего интерес локуса-мишени. В предпочитаемом варианте осуществления изобретения такой модификацией является разрыв цепи.
[25] В таких способах представляющий интерес локус-мишень может являться частью нуклеиновой кислоты in vitro. В таких способах представляющий интерес локус-мишень может являться частью нуклеиновой кислоты, находящейся в клетке. В способе по настоящему изобретению предпочтительно, чтобы представляющий интерес локус-мишень являлся частью молекулы РНК in vitro. В таких способах также предпочтительно, чтобы представляющий интерес локус-мишень являлся частью молекулы РНК, находящейся в клетке. Такая клетка может быть прокариотической или эукариотической. Такая клетка может быть клеткой млекопитающего. Такая клетка может быть клеткой грызуна. Такая клетка может быть клеткой мыши.
[26] В любом из описанных способов представляющий интерес локус-мишень может быть геномным или эпигеномным представляющим интерес локусом. В любом из описанных способов такой комплекс может быть доставлен при помощи многих направляющих молекул для мультиплексированного использования. В каждом из описанных способов может быть использовано более одного белка.
[27] В следующих аспектах изобретения компоненты, являющиеся нуклеиновыми кислотами, могут включать предполагаемую последовательность РНК CRISPR (cr-РНК). Не ограничиваясь этим, заявители предполагают, что в таких случаях пре-cr-РНК может содержать вторичную структуру, обеспечивающую как процессинг с образованием зрелой cr-РНК, так и загрузку cr-РНК в эффекторный белок. В качестве неограничивающего примера, такая вторичная структура может содержать, по существу состоять или состоять из шпилечной структуры в пре-cr-РНК, в частности в составе прямого повтора.
[28] В любом из описанных способов эффекторный белок и компоненты, являющиеся нуклеиновыми кислотами, могут быть предоставлены посредством одной или более полинуклеотидных молекул, кодирующих белок и/или компонент(ы), являющийся нуклеиновой кислотой, причем одна или более полинуклеотидных молекул функционально приспособлены для экспрессии белка и/или компонента(ов), являющегося нуклеиновой кислотой. Одна или более полинуклеотидных молекул могут содержать один или более регуляторных элементов, функционально приспособленных для экспрессии белка и/или компонента(ов), являющегося нуклеиновой кислотой. Одна или более полинуклеотидных молекул может находиться в одном или более векторах. В любом из описанных способов представляющий интерес локус-мишень может быть геномным или эпигеномным представляющим интерес локусом. В любом из описанных способов комплекс может быть доставлен с использованием множественных направляющих молекул для мультиплексированного использования. В любом из описываемых способов может быть использовано более одного белка.
[29] В любом из описанных способов разрыв цепи может быть одноцепочечным или двухцепочечным разрывом.
[30] Регуляторные элементы могут включать индуцибельные промоторы. Полинуклеотиды и/или векторные системы могут являться индуцибельными системами.
[31] В любом из описанных способов одна или более полинуклеотидных молекул могут быть включены в систему доставки, или один или более векторов могут быть включены в систему доставки.
[32] В любом из описанных способов такую не встречающуюся в природе или сконструированную способами инженерии композицию можно доставлять при помощи липосом, и других частиц, включая наночастицы, экзосомы, микровезикулы, генные пушки или одного или более вирусных векторов.
[33] Также изобретение относится к не встречающейся в природе или сконструированной способами инженерии композиции, которая представляет собой композицию, имеющую характеристики, описанные в настоящем описании или определенные в любом из описанных в настоящем описании способов.
[34] Таким образом, в некоторых вариантах осуществления изобретение относится к встречающейся в природе или сконструированной способами инженерии композиции, в частности, композиции, способной или приспособленной для модификации представляющего интерес локуса-мишени, причем указанная композиция включает эффекторный белок локусов CRISPR-Cas типа VI и один или более компонентов, являющихся нуклеиновыми кислотами, причем такой эффекторный белок формирует комплекс с одним или более компонентами, являющимися нуклеиновыми кислотами, и при связывании указанного комплекса с представляющим интерес локусом такой эффекторный белок индуцирует модификацию представляющего интерес локуса-мишени. В некоторых вариантах осуществления изобретения, такой эффекторный белок может быть эффекторным белком локусов С2с2.
[35] Также в следующем аспекте настоящее изобретение относится к не встречающейся в природе или сконструированной способами инженерии композиции, в частности, композиции, способной или приспособленной для модификации представляющего интерес локуса-мишени, причем указанная композиция включает: (a) молекулу направляющей РНК (или комбинацию молекул направляющей РНК, например, первую молекулу направляющей РНК-гида или вторую молекулу направляющей РНК) или нуклеиновую кислоту, кодирующую молекулу направляющей РНК (или одну или более нуклеиновых кислот, кодирующих комбинацию молекул направляющих РНК); (b) эффекторный белок локусов CRISPR-Cas типа VI или нуклеиновую кислоту, кодирующую эффекторный белок локусов CRISPR-Cas типа VI. В некоторых вариантах осуществления такой эффекторный белок может быть эффекторным белком локуса С2с2.
[36] Еще один аспект изобретения также относится к не встречающейся в природе или сконструированной способами инженерии композиции, которая: (a) содержит молекулу направляющей РНК (или комбинацию молекул направляющей РНК, например, первую молекулу направляющей РНК и вторую молекулу направляющей РНК) или нуклеиновую кислоту, кодирующую комбинацию молекул направляющих РНК; (b) является эффекторным белком локуса C2c2.
[37] Также изобретение относится к векторной системе, включающей один или более векторов, причем один или более векторов содержат одну или более полинуклеотидных молекул, кодирующих компоненты с не встречающейся в природе или сконструированной способами инженерии композицией, которая представляет собой композицию, обладающую характеристиками, которые определены в любом из описанных в настоящем описании способов.
[38] Также изобретение относится к системе доставки, содержащей один или более векторов или одну или более полинуклеотидных молекул, причем один или более векторов или полинуклеотидных молекул включают одну или более полинуклеотидных молекул, кодирующих компоненты не встречающейся в природе или сконструированной способами инженерии композиции, которая представляет собой композицию, имеющую характеристики, описанными в настоящем описании или определенными в любом из описанных способов.
[39] Также изобретение относится к не встречающейся в природе или сконструированной способами инженерии композиции, или одному или более полинуклеотидам, кодирующим компоненты указанной композиции, или к вектору либо системе доставки, содержащим один или более полинуклеотидов, кодирующих компоненты указанной композиции для использования в терапевтическом способе лечения. Такой терапевтический способ лечения может включать генное или транскриптомное редактирование или генную терапию.
[40] Также изобретение относится к вычислительным (биоинформатическим) способам и алгоритмам прогнозирования новых систем CRISPR-Cas 2 класса и идентификации их компонентов.
[41] Также изобретение относится к способам и композициям, в которых один и несколько аминокислотных остатков эффекторного белка могут быть модифицированы, например, в сконструированном способами инженерии или не встречающемся в природе эффекторном белке или C2c2. В одном из вариантов осуществления изобретения модификация может включать мутацию одного или более аминокислотных остатков эффекторного белка. Одна или более мутаций могут быть локализованы в одном или более каталитических доменах эффекторного белка. Нуклеазная активность такого эффекторного белка может быть снижена или отсутствовать по сравнению с эффекторным белком, не имеющим указанную одну или более мутаций. Такой эффекторный белок может быть не способен направлять расщепление цепи ДНК или РНК в представляющем интерес локусе-мишени. Предпочтительный вариант осуществления включает две таких мутаций. В предпочтительном варианте осуществления изобретения такие один или более аминокислотных остатков модифицированы в эффекторном белке С2с2, например, в сконструированном способами инженерии или не встречающемся в природе эффекторном белке или C2c2. В некоторых вариантах осуществления изобретения такие один или более модифицированных либо имеющих мутации аминокислотных остатков находятся в C2c2 и соответствуют R597, H602, R1278 и H1283 (с отсылкой к аминокислотам в Lsh C2c2 и консенсусной нумерации C2c2), включая R597A, H602A, R1278A и HI283A, или соответствуют аминокислотным остаткам в ортологах Lsh C2c2.
[42] В конкретных вариантах осуществления изобретения, такие один или более модифицированных или мутантных аминокислотных остатков являются модифицированными аминокислотными остатками C2c2, соответствующими K2, КЗ9, V40, E479, L514, V518, N524, G534, K535, E580, L597, V602, D630, F676, L709, I713, R717 (HEPN), N718, H722 (HEPN), E773, P823, V828, I879, Y880, F884, Y997, L1001, F1009, L1013, Y1093, L1099, L1111, Y1114, L1203, D1222, Y1244, L1250, L1253, K1261, I1334, L1355, L1359, R1362, Y1366, E1371, R1372, D1373, R1509 (HEPN), H1514 (HEPN), Y1543, D1544, К1546, К1548, V1551, I1558, в соответствии с нумерацией консенсусного C2c2. В определенных вариантах осуществления один или более модифицированных либо мутантных аминокислотных остатков являются одним или более аминокислотными остатками C2c2, соответствующими R717 и R1509. В некоторых аспектах осуществления такие один или более модифицированных либо мутантных аминокислотных остатков являются одним или более аминокислотными остатками C2c2, соответствующими K2, K39, K535, K1261, R1362, R1372, К1546 и K1548. В некоторых аспектах осуществления указанные мутации приводят к тому, что белок имеет измененную или модифицированную активность. В некоторых вариантах осуществления указанные мутации приводят к тому, что белок имеет увеличенную активность. В некоторых вариантах осуществления указанные мутации приводят к тому, что такой белок имеет увеличенную активность, например, за счет увеличенной специфичности. В некоторых вариантах осуществления указанные мутации приводят к тому, что такой белок имеет сниженную активность, например, за счет сниженной специфичности. В некоторых вариантах осуществления указанные мутации приводят к тому, что такой белок не имеет каталитической активности (так называемый "мертвый" C2c2). В одном из вариантов осуществления указанные мутации соответствуют аминокислотным остаткам Lsh C2c2 или соответствующим аминокислотными остаткам белка C2c2 других биологических видов.
[43] Также изобретение относится к одной или более мутациям или к двум или более мутациям в каталитически активном домене эффекторного белка. В некоторых вариантах осуществления изобретения такой каталитически активный домен может являться одним из доменов RuvCI, RuvCII или RuvCIII либо каталитически активным доменом, гомологичным доменам RuvCI, RuvCII и RuvCIII и т.д. или любому соответствующему домену, как описано в любом из описанных в настоящем описании способов. В некоторых вариантах осуществления такие одна или более мутаций могут быть локализованы в каталитически активном домене эффекторного белка, являющемся доменом HEPN, или каталитически активном домене, гомологичном домену HEPN. Такой эффекторный белок может иметь один или более доменов, являющихся сигналом ядерной локализации (NLS). Такие один или более гетерологичных функциональных доменов могут содержать по меньшей мере два или более доменов NLS. Такие один или более доменов NLS могут быть расположены в конце или вблизи конца последовательности эффекторного белка (например, C2c2), а в случае двух и более NLS каждый из этих двух может быть расположен вблизи конца эффекторного белка (например, C2c2). Такие один или более гетерологичных функциональных доменов могут являться одним или более доменами активации трансляции. В других вариантах осуществления такой функциональный домен может являться доменом активации трансляции, например, VP64. Такие один или более функциональных доменов могут являться одним или более доменами репрессии транскрипции. В некоторых вариантах осуществления такой домен репрессии транскрипции является доменом KRAB или доменом SID (например, SID4X). Такие один или более гетерологичных функциональных доменов могут являться одним или более нуклеазными доменами. В предпочтительном варианте осуществления нуклеазный домен является доменом Fok1.
[44] Также изобретение относится к одному или более гетерологичным функциональным доменам следующих типов активности: метилазная активность, деметилазная активность, активация транскрипции, репрессия транскрипции, активность фактора терминации транскрипции, модификация гистонов, нуклеазная активность, расщепление одноцепочечной РНК, расщепление двухцепочечной РНК, расщепление одноцепочечной ДНК, расщепление двухцепочечной ДНК и связывание нуклеиновых кислот. По меньшей мере один или более гетерологичных функциональных доменов могут находиться в N-конце эффекторного белка или вблизи него, при этом также по меньшей мере один или более гетерологичных функциональных доменов могут находиться в C-конце эффекторного белка или вблизи него. Такой один или более гетерологичных функциональных доменов могут быть слиты с таким эффекторным белком. Такие один или более гетерологичных функциональных доменов могут быть присоединены к такому эффекторному белку. Такие один или более гетерологичных функциональных доменов могут быть соединены с таким эффекторным белком посредством связующей группы.
[45] Также изобретение относится к эффекторному белку, включающему эффекторный белок из организма, относящегося к одному из следующих родов: Streptococcus, Campylobacter, Nitratifractor, Staphylococcus, Parvibaculum, Roseburia, Neisseria, Gluconacetobacter, Azospirillum, Sphaerochaeta, Lactobacillus, Eubacterium, Corynebacter, Carnobacterium, Rhodobacter, Listeria, Paludibacter, Clostridium, Lachnospiraceae, Clostridiaridium, Leptotrichia, Francisella, Legionella, Alicyclobacillus, Methanomethyophilus, Porphyromonas, Prevotella, Bacteroidetes, Helcococcus, Letospira, Desulfovibrio, Desulfonatronum, Opitutaceae, Tuberibacillus, Bacillus, Brevibacilus, Methylobacterium или Acidaminococcus. Такой эффекторный белок может представлять собой химерный эффекторный белок, первый фрагмент которого получен из первого ортолога эффекторного белка, а второй фрагмент - из второго ортолога эффекторного белка, причем первый и второй ортологи эффекторного белка различаются. По меньшей мере один из первого и второго ортологов эффекторного белка может представлять собой эффекторный белок одного из следующих организмов: Streptococcus, Campylobacter, Nitratifractor, Staphylococcus, Parvibaculum, Roseburia, Neisseria, Gluconacetobacter, Azospirillum, Sphaerochaeta, Lactobacillus, Eubacterium, Corynebacter, Carnobacterium, Rhodobacter, Listeria, Paludibacter, Clostridium, Lachnospiraceae, Clostridiaridium,Leptotrichia, Francisella, Legionella, Alicyclobacillus, Methanomethyophilus, Porphyromonas, Prevotella, Bacteroidetes, Helcococcus, Letospira, Desulfovibrio, Desulfonatronum, Opitutaceae, Tuberibacillus, Bacillus, Brevibacillus, Methylobacterium или Acidaminococcus.
[46] В некоторых вариантах осуществления изобретения такой эффекторный белок, в частности, эффекторный белок локусов типа V, а более конкретно эффекторный белок локусов типа V-B, в частности C2c1p, может происходить из, быть получен из или являться производным метаболизма бактерий, относящегося к таким таксонам как Bacilli, Verrucomicrobia, альфа-протеобактерии или дельта-протеобактерии. В некоторых вариантах осуществления такой эффекторный белок, в частности, эффекторный белок локусов типа V, более конкретно эффекторный белок локусов V-B типа, в частности C2c1p, может происходить из, быть выделен из или являться производным метаболизма бактерий, относящихся к одному из следующих родов: Alicyclobacillus, Desulfovibrio, Desulfonatronum, Opitutaceae, Tuberibacillus, Bacillus, Brevibacillus, Desulfatirhabdium, Citrobacter, и Methylobacterium. В некоторых вариантах осуществления такой эффекторный белок, в частности, эффекторный белок локусов типа V, более конкретно эффекторный белок локусов типа V-B, в частности C2c1p, может происходить из, быть выделен из или являться производным метаболизма следующих бактерий: Alicyclobacillusacidoterrestris (например, ATCC 49025), Alicyclobacilluscontaminans (например, DSM 17975), Desulfovibrioinopinatus (например, DSM 10711), Desulfonatronumthiodismutans (например, штамм MLF-1), Opitutaceaebacterium TAV5, Tuberibacilluscalidus (например, DSM 17572), Bacillusthermoamylovorans (например, штамм B4166), Brevibacillussp. CF112, Bacillussp. NSP2.1, Desulfatirhabdiumbutyrativorans (например, DSM 18734), Alicyclobacillusherbarius (например, DSM 13609), Citrobacterfreundii (например, ATCC 8090), Brevibacillusagri (например, BAB-2500), Methylobacteriumnodulans (например, ORS 2060). В некоторых вариантах осуществления такой эффекторный белок, в частности, эффекторный белок локусов типа V, более конкретно эффекторный белок локусов типа V-B, в частности C2c1p, может происходить из, быть выделен из являться производным метаболизма бактерий из перечня, приведенного в таблице на фиг. 41A-B.
[47] В некоторых вариантах осуществления такой эффекторный белок, а именно белок локусов типа V, более конкретно эффекторный белок локусов типа V-B, в частности C2c1p, может содержать, по существу состоять или состоять из аминокислотной последовательности, выбранной из перечня, включающего аминокислотные последовательности, представленные на множественном выравнивании последовательностей на фиг. 13D-H.
[48] В некоторых вариантах осуществления локус типа V-B, как подразумевается в настоящем описании, может кодировать слитую конструкцию Cas1-Cas4, Cas2 и эффекторный белок C2c1p. В некоторых вариантах осуществления локус типа V-B, как подразумевается в настоящем описании, может примыкать к последовательности CRISPR. Характерная организация локусов типа V-B проиллюстрирована на фиг. 9 и фиг. 41A-B.
[49] В некоторых вариантах осуществления белок Cas1, кодируемый локусом типа V-B, как подразумевается в настоящем описании, может группироваться с системой типа I-U. На фиг. 10A и 10B и 10C-V проиллюстрировано дерево Cas1, включая Cas1, кодируемый репрезентативными локусами типа V-B.
[50] В определенных вариантах осуществления изобретения эффекторный белок, конкретно эффекторный белок локусов типа V, более конкретно эффекторный белок локусов типа V-B, а частности - C2c1p, например, природный C2c1p, может иметь длину от приблизительно 1100 до приблизительно 1500 аминокислот, например, от приблизительно 1100 до приблизительно 1200, или от приблизительно 1200 до приблизительно 1300 аминокислот, или от приблизительно 1300 до приблизительно 1400 аминокислот, или от приблизительно 1400 до приблизительно 1500 аминокислот, например, приблизительно 1100, приблизительно 1200, приблизительно 1300, приблизительно 1400, или приблизительно 1500 аминокислот.
[51] В некоторых вариантах осуществления изобретения эффекторный белок, конкретно эффекторный белок локусов типа V, более конкретно эффекторный белок локусов типа V-B, в частности - C2c1p, и предпочтительно C-концевая часть указанного эффекторного белка, содержит три каталитических мотива RuvC-подобной нуклеазы (т.е. RuvCI, RuvCII и RuvCIII). В некоторых вариантах осуществления изобретения указанный эффекторный белок и предпочтительно С-концевая часть указанного эффекторного белка может далее содержать область, соответствующую мостиковой спирали (также известной как богатый аргинином кластер), которая в белке Cas9 участвует в связывании cr-РНК. В некоторых вариантах осуществления изобретения указанный эффекторный белок и предпочтительно C-концевая часть указанного белка может далее включать домен, содержащий цинковый палец, который может быть неактивным (т.е. который не связывает цинк, например, в котором Zn-связывающие остатки цистеина отсутствуют). В некоторых вариантах осуществления изобретения указанный эффекторный белок и предпочтительно C-концевая часть указанного эффекторного белка может содержать три каталитических мотива RuvC-подобной нуклеазы (т.е. RuvCI, RuvCII и RuvCIII), область, соответствующую мостиковой спирали, домен, содержащий цинковый палец, предпочтительно в следующем порядке, от N- к C-концу: RuvCI-мостиковая спираль-RuvCII-цинковый палец-RuvCIII См. Фиг. 11, фиг. 12 и фиг. 13A и 13C для иллюстрации доменной архитектуры представителей эффекторных белков типа V-B.
[52] В некоторых вариантах осуществления изобретения локусы типа V-B, как описано в настоящем описании, могут содержать CRISPR-повторы длиной от 30 до 40 п.н., более типично от 34 до 38 п.н., еще более типично - от 36 до 37 п.н., например, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39 или 40 п.н.
[53] В некоторых вариантах осуществления изобретения эффекторный белок, конкретно эффекторный белок локусов типа V, более конкретно - эффекторный белок локусов типа V-C, в частности - C2c3p может происходить, быть изолирован или получен из бактериального метагенома, выбранного из бактериальных метагеномов, перечисленных в таблице на фиг. 43 A-B.
[54] В некоторых вариантах осуществления изобретения эффекторный белок, конкретно эффекторный белок локусов типа V, более конкретно эффекторный белок локусов типа V-C, в частности - C2c3p, может включать, состоять в основном из или состоять только из аминокислотной последовательности, выбранной из группы аминокислотных последовательностей, показанных на множественном выравнивании последовательностей на фиг. 131.
[55] В некоторых вариантах осуществления изобретения локус типа V-C, как описано в настоящем описнаии, может кодировать эффекторный белок Cas1 и C2c3p. См. Фиг. 14 и фиг. 43A-B для иллюстрации организации характерных локусов типа V-С.
[56] В некоторых вариантах осуществления изобретения белок Cas1, кодируемый локусом типа V-С, как описано в настоящем описании, может образовывать кластеры с системой типа I-B. См. Фиг. 10A и 10B и фиг. 10C-W, иллюстрирующие дерево Cas-белков, включающее белок Cas1, кодируемый характерными локусами типа V-С.
[57] В некоторых вариантах осуществления изобретения эффекторный белок, а именно белок локусов типа V, более конкретно эффекторный белок локусов типа V-С, еще более конкретно - C2c3p, в частности C2c3p, может иметь длину от приблизительно 1100 до приблизительно 1500 аминокислот, например, от приблизительно 1100 до приблизительно 1200 аминокислот, или от приблизительно 1200 до приблизительно 1300 аминокислот, или от приблизительно 1300 до приблизительно 1400 аминокислот, или от приблизительно 1400 до приблизительно 1500 аминокислот, например, приблизительно 1100, приблизительно 1200, приблизительно 1300, приблизительно 1400 или приблизительно 1500 аминокислот, или как минимум приблизительно 1100, как минимум приблизительно 1200, как минимум приблизительно 1300, как минимум приблизительно 1400 или как минимум приблизительно 1500 аминокислот.
[58] В некоторых вариантах осуществления изобретения эффекторный белок, а именно эффекторный белок локусов типа V, более конкретно эффекторный белок локусов типа V-С, в частности - C2c3p, предпочтительно C-концевая часть указанного эффекторного белка, включает три каталитических мотива RuvC-подобной нуклеазы (т.е., RuvCI, RuvCII и RuvCIII). В некоторых вариантах осуществления изобретения указанный эффекторный белок, предпочтительно C-концевая часть указанного эффекторного белка, может содержать область, соответствующую мостиковой спирали (также известной как богатый аргинином кластер), которая в белке Cas9 участвует в связывании cr-РНК. В некоторых вариантах осуществления изобретения указанный эффекторный белок, предпочтительно C-концевая часть указанного эффекторного белка, может далее включать домен цинкового пальца. Предпочтительно сохранение Zn-связывающих остатков цистеина в C2c3p. В некоторых вариантах осуществления изобретения указанный эффекторный белок, предпочтительно C-концевая часть указанного эффекторного белка, может включать три каталитических мотива RuvC-подобной нуклеазы (т.е., RuvCI, RuvCII и RuvCIII), область, соответствующую мостиковой спирали, и домен цинкового пальца, предпочтительно в следующем порядке с N к C концу: RuvCI-мостиковая спираль-RuvCII-цинковый палец-RuvCIII. См. Фиг. 13A и 13C для иллюстрации характерной доменной архитектуры эффекторных белков типа V-С. В конкретных вариантах осуществления указанный эффекторный белок может включать два HEPN каталитических мотива, как показано на фиг. 97(A).
[59] В некоторых вариантах осуществления изобретения локусы типа V-С, как предполагается в настоящем описании, могут содержать CRISPR-повторы длиной от 20 до 30 п.н., более типично от 22 до 27 п.н. длиной, и еще более типично 25 п.н. длиной, например, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, или 30 п.н.
[60] В некоторых вариантах осуществления изобретения эффекторный белок, а именно эффекторный белок локусов типа VI, в частности C2c2p, может происходить, быть выделенными или полученным из бактерий, принадлежащих к таксонам альфа-протеобактерии, Bacilli, Clostridia, Fusobacteria и Bacteroidetes. В некоторых вариантах осуществления изобретения эффекторный белок, конкретнее эффекторный белок локусов типа VI, в частности - C2c2p, может происходить, быть выделенным или полученным из бактерий, принадлежащих к одному из следующих родов: Lachnospiraceae, Clostridium, Carnobacterium, Paludibacter, Listeria, Leptotrichia и Rhodobacter. В некоторых вариантах осуществления изобретения эффекторный белок, а именно эффекторный белок локусов типа VI, в частности C2c2p, может происходить, быть выделенным или полученным из следующих видов бактерий: Lachnospiraceaebacterium MA2020, Lachnospiraceaebacterium NK4A179, Clostridiumaminophilum (например, DSM 10710), Lachnospiraceaebacterium NK4A144, Carnobacteriumgallinarum (например, DSM 4847 штамм MT44), Paludibacterpropionicigenes (например, WB4), Listeriaseeligeri (например, серовар  штамм SLCC3954), Listeriaweihenstephanensis (например, FSL R9-0317 c4), Listerianewyorkensis (например, штамм FSL M6-0635), Leptotrichiawadei (например, F0279), Leptotrichiabuccalis (например, DSM 1135), Leptotrichiasp. Oraltaxon 225 (например, str. F0581), Leptotrichiasp. Oraltaxon 879 (например, штамм F0557), Leptotrichiashahii (например, DSM.19757), Rhodobactercapsulatus (например, SB 1003, R121, или DE442). В некоторых вариантах осуществления эффекторный белок, более конкретно эффекторный белок локусов типа V, еще более конкретно - C2c2p может происходить, быть выделенным или полученным из видов бактерий, перечисленных в таблице в фиг. 42А-В. В конкретных вариантах осуществления белок C2c2 происходит из Leptotrichiashahii (например, DSM 19757).
штамм SLCC3954), Listeriaweihenstephanensis (например, FSL R9-0317 c4), Listerianewyorkensis (например, штамм FSL M6-0635), Leptotrichiawadei (например, F0279), Leptotrichiabuccalis (например, DSM 1135), Leptotrichiasp. Oraltaxon 225 (например, str. F0581), Leptotrichiasp. Oraltaxon 879 (например, штамм F0557), Leptotrichiashahii (например, DSM.19757), Rhodobactercapsulatus (например, SB 1003, R121, или DE442). В некоторых вариантах осуществления эффекторный белок, более конкретно эффекторный белок локусов типа V, еще более конкретно - C2c2p может происходить, быть выделенным или полученным из видов бактерий, перечисленных в таблице в фиг. 42А-В. В конкретных вариантах осуществления белок C2c2 происходит из Leptotrichiashahii (например, DSM 19757).
[61] В некоторых вариантах осуществления изобретения эффекторный белок, а именно, эффекторный белок локусов типа VI, в частности - C2c2p, может содержать, состоять только из или включать аминокислотную последовательность, выбранную из последовательностей, показанных на множественном выравнивании на фиг. 13J-N или, более конкретно, состоящих из аминокислотных последовательностей, показанных на выравнивании на фиг. 110.
[62] В некоторых вариантах осуществления изобретения локус типа VI, как предполагается в рамках настоящего изобретения, может кодировать эффекторные белки Cas1, Cas2 и C2c2p. В некоторых вариантах осуществления изобретения локус типа V-С, как предполагается в рамках настоящего изобретения, может содержать последовательность CRISPR. В некоторых вариантах осуществления изобретения локус типа V-С, как предполагается в рамках настоящего изобретения, может содержать ген c2c2 и последовательность CRISPR, и не содержать гены cas1 и cas2. См. Фиг. 15 и фиг. 42A-В для иллюстрации характерной организации локусов типа VI.
[63] В некоторых вариантах осуществления изобретения белок Cas1, кодируемый локусом типа VI, как предполагается в рамках настоящего изобретения, может образовывать кластеры с поддеревом типа II, включая небольшую ветвь типа III-A, или внутри системы типа III-A. См. Фиг. 10A и 10B и фиг. 10C-W, на которых изображено дерево Cas1, включающее белок Cas1, кодируемый типичными локусами типа VI.
[64] В некоторых вариантах осуществления изобретения эффекторный белок, конкретно эффекторный белок локусов типа VI, в частности C2c2p, например природный C2c2p, может быть от приблизительно 1000 до приблизительно 1500 аминокислотных остатков длиной, таким как от приблизительно 1100 до приблизительно 1400 аминокислот длиной, например, от приблизительно 1000 до приблизительно 1100 аминокислот длиной, от приблизительно 1100 до приблизительно 1200 аминокислот длиной, или от приблизительно 1200 до приблизительно 1300 аминокислот длиной, или от приблизительно 1300 до приблизительно 1400 аминокислот длиной, или от приблизительно 1400 до приблизительно 1500 аминокислот длиной, например, приблизительно 1000, приблизительно 1100, приблизительно 1200, приблизительно 1300, приблизительно 1400 или приблизительно 1500 аминокислот длиной.
[65] В некоторых вариантах осуществления изобретения эффекторный белок, а именно эффекторный белок локусов типа VI, а частности C2c2p, содержит как минимум один или предпочтительно как минимум два, наиболее предпочтительно ровно два, консервативных мотива RxxxxH. Каталитические RxxxxH-мотивы характерны для HEPN-доменов (домен, связывающийся с ДНК, присутствующий у эукариот и прокариот). Следовательно, в некоторых вариантах осуществления изобретения эффекторный белок, а именно эффекторный белок локусов типа VI, в частности - C2c2p, содержит как минимум один или предпочтительно как минимум два, наиболее предпочтительно ровно два, HEPN-домена. См. Фиг. 11 и фиг. 13B и фиг. 110A для иллюстрации характерной доменной архитектуры эффекторных белков типа VI. В некоторых вариантах осуществления изобретения HEPN-домены могут обладать РНК-азной активностью. В других вариантах осуществления изобретения HEPN-домены могут обладать ДНК-азной активностью.
[66] В некоторых вариантах осуществления изобретения локусы типа VI, как предполагается в рамках настоящего изобретения, могут содержать CRISPR-повторы длиной от 30 до 40 п.н., более типично длиной от 35 до 39 п.н., например, длиной 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, или 40 п.н. В конкретных вариантах осуществления изобретения длина прямого повтора составляет как минимум 25 п.н.
[67] В некоторых вариантах осуществления изобретения последовательность, прилегающая к протоспейсеру (PAM), или PAM-подобный мотив управляет связыванием эффекторного белкового комплекса с представляющим интерес локусом-мишенью, как описано настоящем описании. В некоторых вариантах осуществления изобретения, PAM может представлять собой 5'-PAM (т.е. быть расположенным выше 5'-конца протоспейсера). В других вариантах осуществления изобретения PAM может представлять собой 3'-PAM (т.е. быть расположенным ниже 5'-конца протоспейсера). Термин "PAM" может быть использован взаимозаменяемо с термином "PFS" или "участок, фланкирующий протоспейсер" или "последовательность, фланкирующая протоспейсер".
[68] В предпочтительном варианте осуществления изобретения эффекторный белок, конкретно эффекторный белок локусов типа V, более конкретно эффекторный белок локусов типа V-B, еще более конкретно - C2c1p, может распознавать 5'-PAM. В некоторых вариантах осуществления изобретения эффекторный белок, конкретно эффекторный белок локусов типа V, более конкретно - эффекторный белок локусов типа V-B, еще более конкретно - C2c1p, может распознавать 5'-PAM, который является 5'-TTN- или 5'-ATTN-последовательностью, где N - это A, C, G или T. В некоторых предпочтительных вариантах осуществления изобретения эффекторный белок может быть белком C2c1p бактерии Alicyclobacillus acidoterrestris, более предпочтительно - белком ATCC 49025 C2c1p бактерии Alicyclobacillus acidoterrestris, в котором 5'-PAM представлен 5'-последовательностью TTN, где N - это A, C, G или T, более предпочтительно где N - это A, G или T. В других наилучших вариантах осуществления изобретения эффекторный белок является белком C2c1p бактерии Bacillus thermoamylovorans, более предпочтительно - белком C2c1p штамма B4166 бактерии Bacillusthermo amylovorans, в котором 5'-PAM представлен 5'-последовательностью ATTN, где N - это A, C, G или T.
[69] В предпочтительном варианте осуществления изобретения эффекторный белок, конкретно эффекторный белок локусов типа VI, более конкретно - C2c2p, может распознавать 3'-PAM. В некоторых вариантах осуществления изобретения эффекторный белок, а именно - эффекторный белок локусов типа VI, в частности - C2c2p, может распознавать 3'-PAM, который представлен 5'-H, где H - это A, C или U. В некоторых предпочтительных вариантах осуществления изобретения эффекторный белок может быть белком C2c2p бактерии Leptotrichia shahii, более предпочтительно - белком DSM 19757 C2c2 бактерии Leptotrichia shahii, в котором 5'-PAM представлен 5'-H.
[70] В некоторых вариантах осуществления изобретения фермент CRISPR сконструирован способами инженерии и может содержать одну и несколько мутаций, которые уменьшают или полностью устраняют нуклеазную активность. Мутации также могут быть внесены в соседние остатки, например, в аминокислоты возле указанных выше, которые обеспечивают нуклеазную активность. В некоторых вариантах осуществления изобретения только один HEPN-домен инактивирован, в других вариантах, второй HEPN-домен тоже инактивирован.
[71] В некоторых вариантах осуществления изобретения направляющая РНК или зрелая cr-РНК содержит, состоит в основном из или состоит только из прямой повторяющейся последовательности и направляющей последовательности или спейсера. В некоторых вариантах осуществления изобретения направляющая РНК или зрелая cr-РНК состоит из частичного прямого повтора размером 19 нуклеотидов с последующей направляющей последовательностью размером 18, 19, 20, 21, 22, 23, 24, 25, или более нуклеотидов, например, направляющей последовательностью или спейсером размером 18-25, 19-25, 20-25, 21-25, 22-25 или 23-25 нуклеотидов. В некоторых вариантах осуществления эффекторный белок представлен эффекторным белком C2c2 и требует направляющей последовательности длиной как минимум 16 нуклеотидов для достижения поддающегося обнаружению расщепления ДНК и направляющей последовательности размером минимум 17 нуклеотидов для достижения эффективного расщепления ДНК in vitro. В конкретных вариантах осуществления изобретения эффекторный белок представлен белком C2c2 и требует направляющей последовательности размером как минимум 19 нуклеотидов для достижения поддающегося обнаружению расщепления РНК. В некоторых вариантах осуществления прямая повторяющаяся последовательность расположена выше (т.е. на 5'-конце) направляющей последовательности или спейсера. В предпочтительном варианте осуществления последовательность затравки (т.е. последовательность, необходимая для распознавания и/или гибридизации с последовательностью локуса-мишени) направляющей РНК C2c2 находится примерно в пределах первых 5 нуклеотидов на 5'-конце направляющей последовательности или спейсера.
[72] В предпочтительных вариантах осуществления изобретения зрелая cr-РНК содержит шпилечную структуру или оптимизированную шпилечную структуру или любую оптимизированную вторичную структуру. В предпочтительных вариантах осуществления изобретения зрелая cr-РНК содержит шпилечную структуру или оптимизированную шпилечную структуру в прямой повторяющейся последовательности, где шпилечная структура или оптимизированная шпилечная структура важна для расщепления. В некоторых вариантах осуществления изобретения зрелая cr-РНК предпочтительно включает единичную шпилечную структуру. В некоторых вариантах осуществления изобретения прямая повторяющаяся последовательность предпочтительно включает единичную шпилечную структуру. В некоторых вариантах осуществления изобретения активность расщепления комплекса эффекторного белка может быть модифицирована путем внесения мутаций, которые влияют на структуру дуплекса РНК шпилечной структуры. В предпочтительных вариантах осуществления изобретения могут быть внесены мутации, поддерживающие структуру дуплекса РНК шпилечной структуры, благодаря которым будет сохранятся ферментативная активность, осуществляемая комплексом эффекторного белка. В других предпочтительных вариантах осуществления изобретения мутации, нарушающие структуру дуплекса РНК шпилечной структуры, могут быть внесены для полного удаления ферментативной активности, осуществляемой комплексом эффекторного белка.
[73] В конкретных вариантах осуществления изобретения белок C2c2 представляет собой эффекторный белок Lsh C2c2, и зрелая cr-РНК включает структуру или оптимизированную шпилечную структуру. В конкретных вариантах осуществления изобретения прямой повтор cr-РНК содержит по меньшей мере 25 нуклеотидов, включая шпилечную структуру. В конкретных вариантах осуществления изобретения шпилька подвержена заменам отдельных оснований, но активность нарушается только при изменении большей части вторичной структуры или укорочении cr-РНК. Примеры нарушающих активность мутаций включают замену более двух из нуклеотидов шпильки, добавление непарных нуклеотидов к шпильке, сокращение шпильки (удаление одного из образующих пару нуклеотидов) или удлинение шпильки (добавление одной пары нуклеотидов). Однако cr-РНК может быть подвергнута 5'- и/или 3'-удлинению, чтобы включать нефункциональные последовательности РНК, как предусмотрено для конкретных применений, описанных в настоящем описании.
[74] Также изобретение относится к нуклеотидной последовательности, кодирующей эффекторный белок, являющейся кодон-оптимизированной для экспрессии в эукариотическом организме или эукариотической клетке посредством любого из описанных в настоящем описании способов или композиций. В одном варианте осуществления изобретения кодон-оптимизированная нуклеотидная последовательность, кодирующая эффекторный белок, кодирует любой C2c2, обсуждаемый в настоящем описании, и подвергнута оптимизации кодонов для удобства использования в эукариотической клетке или организме, например, такой клетке или организме, как описано в настоящем описании в иных местах, например, но не ограничиваясь этим, в клетке дрожжей, или клетке или организме млекопитающего, включая клетку мыши, клетку крысы, и клетку человека или не являющегося человеком эукариотического организма, например, растения.
[75] В некоторых вариантах осуществления изобретения по меньшей мере один сигнал ядерной локализации (NLS) присоединен к последовательностям нуклеиновых кислот, кодирующим эффекторные белки C2c2. В предпочтительных вариантах осуществления изобретения по меньшей мере одна или более C-концевых или N-концевых последовательностей NLS присоединены (следовательно, молекулы нуклеиновых кислот, кодирующие эффекторный белок C2c2, могут включать код для последовательностей NLS, чтобы экспрессируемый продукт имел присоединенную последовательность NLS). В некоторых вариантах осуществления изобретения по меньшей мере один сигнал ядерного экспорта (NES) присоединен к последовательностям нуклеиновых кислот, кодирующим эффекторные белки C2c2. В предпочтительных вариантах осуществления изобретения по меньшей мере одна или более C-концевых или N-концевых последовательностей NES присоединены (следовательно, молекулы нуклеиновых кислот, кодирующие эффекторный белок C2c2, могут включать код для NES-последовательностей, чтобы экспрессируемый продукт имел присоединенную последовательность NES). В предпочтительном варианте осуществления изобретения C-концевые и/или N-концевые NLS или NES присоединены для оптимальной экспрессии и нацеливания в ядро эукариотических клеток, предпочтительно клеток человека. В предпочтительном варианте осуществления изобретения кодон-оптимизированным эффекторным белком является C2c2 и длина спейсера направляющей РНК составляет от 15 до 35 нуклеотидов. В некоторых вариантах осуществления изобретения длина спейсера направляющей РНК - по меньшей мере 16 нуклеотидов, такая как по меньшей мере 17 нуклеотидов, предпочтительно по меньшей мере 18 нуклеотидов, такая как предпочтительно по меньшей мере 19 нуклеотидов, по меньшей мере 20 нуклеотидов, по меньшей мере 21 нуклеотид или по меньшей мере 22 нуклеотида. В некоторых вариантах осуществления изобретения длина спейсера составляет от 15 до 17 нуклеотидов, от 17 до 20 нуклеотидов, от 20 до 24 нуклеотидов, например, 20, 21, 22, 23 или 24 нуклеотидов, от 23 до 25 нуклеотидов, например, 23, 24 или 25 нуклеотидов, от 24 до 27 нуклеотидов, 27-30 нуклеотидов, 30-35 нуклеотидов или 35 нуклеотидов или длиннее. В некоторых вариантах осуществления изобретения кодон-оптимизированным эффекторным белком является C2c2, и длина прямого повтора направляющей РНК составляет по меньшей мере 16 нуклеотидов. В некоторых вариантах осуществления изобретения кодон-оптимизированным эффекторным белком является C2c2, и длина прямого повтора направляющей РНК составляет от 16 до 20 нуклеотидов, например, 16, 17, 18, 19 или 20 нуклеотидов. В определенных предпочтительных вариантах осуществления изобретения длина прямого повтора направляющей РНК равна 19 нуклеотидам.
[76] Настоящее изобретение также охватывает способы для доставки множественных компонентов, являющихся нуклеиновыми кислотами, где каждый компонент, являющийся нуклеиновой кислотой, специфичен к различным представляющим интерес локусам-мишеням для изменения множественных представляющих интерес локусов-мишеней. Компонент, являющийся нуклеиновой кислотой, комплекса может включать один или более связывающих белок РНК-аптамеров. Один или более аптамеров могут быть способны к связыванию с белком оболочки бактериофага. Белок оболочки бактериофага может быть выбран из группы, включающей: Qβ, F2, GA, fr, JP501, MS2, M12, R17, BZ13, JP34, JP500, KU1, М11, MX1, TW18, VK, SP, FI, ID2, NL95, TW19, AP205, ϕCb5, ϕCb8r, ϕCb12r, ϕCb23r, 7s и PRR1. В предпочтительном варианте осуществления изобретения белок оболочки бактериофага представлен белком MS2. Изобретение также предусматривает, что компонент, являющийся нуклеиновой кислотой, комплекса имеет длину 30 или больше, 40 или больше или 50 или больше нуклеотидов.
[77] Соответственно, задачей изобретения не является охватить в своих рамках какой-либо ранее известный продукт, процесс получения продукта или способ применения, таким образом, заявители сохраняют за собой право и настоящим подтверждают отказ от какого-либо ранее известного продукта, процесса или способа. Далее отмечено, что изобретение не намеревается охватить в своих рамках какой-либо продукт, процесс получения или способ применения продукта, которые не соответствует письменному описанию и разрешающим условиям USPTO (35 Свод Законов США 112, параграф первый) или EPO (Статья 83 EPC), так что Заявители сохраняют за собой право и настоящим подтверждают отказ от каого-либо ранее описанного продукта, процесса получения или способа применения продукта. В практике изобретения следует руководствоваться Статьей 53 (c) EPC и Правилом 28 (b) и (c) EPC. Ничто в настоящем описании не может быть истолковано как обещание.
[78] Отмечено, что в настоящей заявке и особенно в формуле изобретения и/или параграфах термины, такие как "содержать", "содержавшийся", "содержащий" и т.п., могут иметь значение, приписанное им в американском Патентном Законе; например, они могут означать "включает", "включенный", "включающий", и т.п.; термины, такие как "по существу состоящий из" и "по существу состоит из" имеют значение, приписанное им в американском Патентном праве, например, они включают элементы, не указанные явно, но исключают элементы, которые учтены в предшествующих заявках или которые влияют на основные или новые характеристики изобретения.
[79] Дальнейшим аспектом изобретения является, что в эукариотической клетке, включающей нуклеотидную последовательность, кодирующую систему CRISPR, описанную в настоящем описании, происходит модификация представляющего интерес локуса-мишени согласно любому из способов, описанных в настоящем описании. Еще один аспект изобретения относится к созданию клеточной линии указанной клетки. Следующий аспект изобретения предусматривает создание многоклеточного организма, включающего одну или более указанных клеток.
[80] В некоторых вариантах осуществления изобретения модификация представляющего интерес локуса-мишени может приводить к появлению эукариотической клетки с измененной экспрессией продукта по меньшей мере одного гена; эукариотической клетки с измененной экспрессией продукта по меньшей мере одного гена, причем экспрессия этого продукта увеличена; эукариотической клетки с измененной экспрессией продукта по меньшей мере одного гена, причем экспрессия этого продукта уменьшена; или эукариотической клетки, содержащей отредактированный геном.
[81] В некоторых вариантах осуществления изобретения эукариотическая клетка может быть клеткой млекопитающего или человека.
[82] В дальнейших вариантах осуществления изобретения не встречающиеся в природе или сконструированные способами инженерии композиции, векторные системы или системы доставки, в соответствии с настоящим описанием, могут использоваться для специфической интерференции последовательности РНК, специфического модулирования экспрессии последовательности РНК (включая специфичную экспрессию изоформ), стабилизации, локализации, функционализации (например, рибосомных РНК или микроРНК), и т.д.; или мультиплексирования таких процессов.
[83] В дальнейших вариантах осуществления изобретения не встречающиеся в природе или сконструированные способами инженерии композиции, векторные системы или системы доставки, как описано в настоящем описании, могут быть использованы для обнаружения РНК и/или определения ее количества в клетке.
[84] В дальнейших вариантах осуществления изобретения не встречающиеся в природе или сконструированные способами инженерии композиции, векторные системы или системы доставки, как описано в настоящем описании, могут быть использованы для создания моделей болезней и/или систем скрининга.
[85] В дальнейших вариантах осуществления изобретения не встречающиеся в природе или сконструированные способами инженерии композиции, векторные системы или системы доставки, как описано в настоящем описании, могут использоваться для сайт-специфического редактирования или изменения транскриптома; специфической интерференции последовательностей нуклеиновых кислот; или мультиплексной инженерии генома.
[86] Также изобретение относится к получению генного продукта из клетки, клеточной линии или организма, как описано в настоящем описании. В некоторых вариантах осуществления изобретения количество экспрессированного генного продукта может быть больше или меньше, чем количество генного продукта в клетке без измененной экспрессии или отредактированного генома. В некоторых вариантах осуществления изобретения генный продукт может быть изменен по сравнению с генным продуктом из клетки без измененной экспрессии или отредактированного генома.
[87] Эти и другие варианты осуществления изобретения раскрыты в, очевидны из или охвачены следующим Подробным Описанием.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
[88] Новые признаки изобретения подробно сформулированы в приложенной формуле изобретения. Лучшее понимание признаков и преимуществ данного изобретения будет достигнуто при ознакомлении со следующим подробным описанием, которое включает материалы, иллюстрирующие принципы изобретения, и сопроводительные Фиг. к ним:
[89] На фиг. 1A-1B изображена новая классификация систем CRISPR-Cas. Класс 1 включает многосубъединичные эффекторные комплексы cr-РНК (Cascade), и Класс 2 включает односубъединичные эффекторные комплексы cr-РНК (Cas9-подобные). На фиг. 1B приведено другое описание новой классификации систем CRISPR-Cas.
[90] На фиг. 2 изображена молекулярная организация систем CRISPR-Cas.
[91] На фиг. 3A-3D изображены структуры эффекторных комплексов типов I и III: общая архитектура/общее происхождение прослеживается несмотря на обширную дивергенцию последовательностей.
[92] На фиг. 4 изображена CRISPR-Cas как система, основанная на мотивах распознавания РНК (RRM).
[93] На фиг. 5 изображена филогению Cas1, где показан главный аспект эволюции системы CRISPR-Cas - рекомбинация эффекторных модулей cr-РНК в целях адаптации.
[94] На фиг. 6 представлен перечень систем CRISPR-Cas, а именно распространение типов/подтипов систем CRISPR-Cas среди архей и бактерий.
[95] На фиг. 7 изображен алгоритм для идентификации Cas-кандидатов.
[96] На фиг. 8A-8B изображена организация полных локусов систем CRISPR-Cas класса 2. Обозначены три подтипа типа II и подтипы V-A, V-B и V-C и тип VI. Подсемейства на основе филогении Cas1 также обозначены. Схема включает только общие гены, представленные в каждом подтипе; дополнительные гены, присутствующие в некоторых вариантах, опущены. Красным прямоугольником обозначен вырожденный повтор. Серые стрелки показывают направление транскрипции последовательности CRISPR. PreFran, Prevotella-Francisella. На фиг. 8B приведено другое описание организации полных локусов нескольких систем CRISPR-Cas класса 2.
[97] На фиг. 9 изображены окрестности C2c1, т.е. геномная архитектура локусов C2c1 CRISPR-Cas. Обозначено количество повторов в последовательностях CRISPR. Для каждого геномного контига обозначен числовой ID Genebank и координаты локуса.
[98] На фиг. 10A-10B. Фиг. 10A и 10B изображены представления о дереве последовательностей белка Cas1. Дерево на фиг. 10B было построено на основе множественного выравнивания 1498 последовательностей Cas1, которые содержали 304 филогенетически информативных позиции. Ветви, соответствующие системам класса 2 подчеркнуты: голубым - тип II; оранжевым - подтип V-A; красным - подтип V-B; коричневым - подтип V-C; фиолетовым - тип VI. Вставки показывают расширенные ветви новых (под)типов. Значения поддержки бутстрэпа приведены в процентах и показаны только для некоторых соответствующих ветвей.
[99] На рисунках 10C-10W показано полное дерево Cas1, которое схематично показано на фиг. 10B в формате Newick с видовыми названиями и значениями поддержки бутстрэпа. Дерево было построено с помощью программы FastTree (опции "-gamma -wag"). Множественное выравнивание последовательностей Cas1 было отфильтровано с порогом однородности 0,1 и порогом возникновения промежутка 0,5 до реконструкции дерева.
[100] На фиг. 11 изображена доменная организация семейств класса 2.
[101] На фиг. 12 изображены участки гомологии TnpB белков класса 2.
[102] Фиг. 13A-13N. Фиг. 13A и 13B содержат другое изображение доменной архитектуры и консервативных мотивов эффекторных белков класса 2. Фиг. 13A иллюстрирует типы II и V: нуклеазы-производные TnpB. Верхний ряд на рисунке демонстрирует нуклеазы RuvC из Thermos thermophilus (PDB ID: 4EP5) с обозначением каталитических аминокислотных остатков. Ниже показана архитектура каждого домена, приведено выравнивание консервативных мотивов для отдельных представителей соответствующих семейств белков (единственная последовательность для RuvC). Каталитические остатки показаны белыми буквами на черном фоне, консервативные гидрофобные остатки выделены желтым; консервативные остатки малых аминокислот выделены зеленым; в выравнивании спирального мостика положительно заряженные остатки выделены красным. Предсказанная вторичная структура показана ниже выровненных последовательностей: H обозначает α-спирали, Е обозначает выпрямленную конформацию (β-структура). Малоконсервативные последовательности спейсеров между блоками выравнивания обозначены числами. Фиг. 13B иллюстрирует тип VI: белки содержат 2 HEPN-домена, которые могут демонстрировать РНК-азную активность. Верхние блоки выравнивания содержат отдельные описанные ранее домены HEPN, нижние блоки содержат каталитические мотивы из эффекторных белков типа VI. Обозначения как на фиг. 13 A. Фиг. 13C иллюстрирует ближайшие гомологи новых эффекторных белков типа V из числа кодируемых транспозонами белков: непересекающиеся наборы гомологов. Фиг. 13D-H иллюстрирует множественное выравнивание семейства белка C2c1. Данное выравнивание выполнено с использованием программы MUSCLE и изменено вручную на основе локальных попарных выравниваний, полученных с помощью PSI-BLAST. Для каждой последовательности приведен номер-идентификатор GenBank (GI) и систематическое название организма. Вторичная структура предсказана с помощью Jpred и показана ниже последовательности, использованной в качестве последовательности-запроса (обозначения: H - альфа-спираль, E - бета-структура). CONSENSUS рассчитан для каждого столбца выравнивания путем сравнения суммы парных весов каждого столбца для гомогенных столбцов (совпадающий остаток во всех выровненных последовательностях) и случайного столбца с порогом гомогенности 0,8. Мотивы активного сайта RuvC-подобного домена показаны ниже выравнивания. На фиг. 13I представлено множественное выравнивание для семейства белка C2c3. Данное выравнивание выполнено с использованием программы MUSCLE. Для каждой последовательности обозначены внутренний приписанный ей номер и номер-идентификатор GenBank (GI) метагеномного контига, кодирующего соответствующий белок С2с3. Вторичная структура предсказана с помощью Jpred и показана ниже выравнивания (обозначения: H - альфа-спираль, E - бета-структура). CONSENSUS рассчитан для каждого столбца выравнивания путем сравнения суммы парных весов каждого столбца для гомогенных столбцов (совпадающие остатки во всех выровненных последовательностях) и случайного столбца с порогом гомогенности 0,8. Мотивы активного сайта RuvC-подобного домена показаны ниже выравнивания С-концевого домена. Фиг. 13J-N иллюстрирует множественное выравнивание семейства белка C2c2. Выравнивание выполнено с использованием программы MUSCLE и изменено вручную на основе локальных выравниваний с помощью PSI-BLAST. Для каждой последовательности обозначен номер-идентификатор GenBank (GI) и систематическое название организма. Вторичная структура предсказана с помощью Jpred и показана ниже последовательности, использованной в качестве последовательности-запроса (обозначения: H - альфа-спираль, E - бета-структура). CONSENSUS рассчитан для каждого столбца выравнивания путем сравнения суммы парных весов каждого столбца для гомогенных столбцов (совпадающие остатки во всех выровненных последовательностях) и случайного столбца с порогом гомогенности 0,8. Мотивы активных сайтов домена HEPN показаны ниже выравнивания.
[103] На фиг. 14 изображены окрестности C2c3, т.е. геномная архитектура C2c3-локусов системы CRISPR-Cas. Указано количество повторов в последовательностях CRISPR. Для каждого геномного контига обозначены номер-идентификатор GenBank и координаты локуса.
[[104] На фиг. 15 изображены окрестности C2c2.
[105] На фиг. 16 изображены мотивы RxxxxH домена HEPN семейства C2c2.
[106] На фиг. 17 изображен C2C1: 1. Alicyclobacillus acidoterrestris ATCC 49025.
[107] На фиг. 18 изображен C2C1: 4. Desulfonatronum thiodismutans штамма MLF-1.
[108] На фиг. 19 изображен C2C1: 5. бактерии Opitutaceae TAV5.
[109] На фиг. 20 изображен C2C1: 7. Bacillus thermoamylovorans штамма B4166.
[110] На фиг. 21 изображен C2C1: 9. Bacillussp. NSP2.1.
[111] На фиг. 22 изображен C2C2: 1. бактерии Lachnospiraceae MA2020.
[112] На фиг. 23 изображен C2C2: бактерии 2. Lachnospiraceae NK4A179.
[113] На фиг. 24 изображен C2C2: 3. [Clostridium] aminophilum DSM 10710.
[114] На фиг. 25 изображен C2C2: 4. бактерии Lachnospiraceae NK4A144.
[115] На фиг. 26 изображен C2C2: 5. Carnobacterium gallinarum DSM 4847.
[116] На фиг. 27 изображен C2C2: 6. Carnobacterium gallinarum DSM 4847
[117] На фиг. 28 изображен C2C2: 7. Paludibacter propionicigenes WB4.
[118] На фиг. 29 изображен C2C2: 8. Listeria seeligeri серовара 1/2b.
[119] На фиг. 30 изображен C2C2: 9. Listeria weihenstephanensis FSL R9-0317.
[120] На фиг. 31 изображен C2C2: 10. Listeria bacterium FSL M6-0635.
[121] На фиг. 32 изображен C2C2: 11. Leptotrichia wadei F0279.
[122] На фиг. 33 изображен C2C2: 12. Leptotrichia wadei F0279.
[123] На фиг. 34 изображен C2C2: 14. Leptotrichia shahii DSM 19757.
[124] На фиг. 35 изображен C2C2: 15. Rhodobacter capsulatus SB 1003.
[125] На фиг. 36 изображен C2C2: 16. Rhodobacter capsulatus R121.
[126] На фиг. 37 изображен C2C2: 17. Rhodobacter capsulatus DE442.
[127] На фиг. 38 изображено дерево DR-последовательностей.
[128] На фиг. 39 изображено дерево белков C2c2.
[129] Фиг. 40A-40D содержит таблицу, включающую описания 63 больших кодирующих белки генов вблизи генов cas1, идентифицированных с помощью описанного в настоящем описании вычислительного способа. Описанные в настоящем описании представители новых подтипов (V-B, V-C, VI) выделены цветом. Последовательности белков для представителей типа V-B и типа IV, кодирующих AUXO014641567.1, AUXO011689375.1, AUXO011689375.1, AIJXO011277409.1, AUXO014986615.1 не проанализированы, т.к. для этих последовательностей не может быть установлена видовая принадлежность.
[130] Фиг. 41A-41M. Фиг. 41A-B содержат таблицу, демонстрирующую результаты анализа локусов V- B типа (кодирующих белок C2c1). * cas1cas4 - ген, содержащий домены cas4 и cas1; CRISPR - CRISPR-повторы; SOS - гены SOS-ответа; unk - гипотетический белок; > - направление кодирующей последовательности гена; [D] - вырожденный повтор (определены, где возможно); [T] - tracr-РНК. Фиг. 41C-J иллюстрирует анализ CRISPR-последовательностей для локусов V-B типа (кодирующих белки C2c1), как описано в настоящем описании (раздел CRISPR является частью базового результата работы pilercr (см. описание результата работы на сайте pilercr: http://www.drive5.com/pHercr/); фолдинг повторов выполнен с помощью mfold (см. описание результата работы на сайте mfold: http://mfold.ma.albany.edu/?q=mfQld/DNA-Folding-FGrm); результаты фолдинга повторов и анализа CRISPR-последовательностей помещены после детального описания каждого случая; расположение CRISPR см. по ссылке в таблице на фиг. 41 A-В. Фиг. 41K иллюстрирует классификацию CRISPRmap CRISPR-повторов локусов типа V-B (кодирующих белок C2c1) как описано в настоящем описании с помощью CRISPRmap (детали см. http://rna.informatik.uni-freiburg.de/CRISPRmap/Input.jsp). Фиг. 41L иллюстрирует вырожденные повторы локусов V-B типа (кодирующие белок C2c1) обнаруженные, как описано в настоящем описании, с помощью алгоритма поиска CRISPR-последовательностей (http://crispr.u-psud.fr/Server/). Столбец нормальных повторов содержит нормальный повтор, столбец спейсеров - последний спейсер, столбец downstream - область низлежащих последовательностей, начиная с вырожденного повтора (250 п.н.); номер последовательности соответствует номеру последовательности CRISPR в соответствующем локусе (см. Таблицу на фиг. 41 A-В); выделенная желтым область имеет полное совпадение между нормальным повтором и вырожденным повтором (при несовпадении с другой частью вырожденного повтора). Фиг. 41M иллюстрирует предсказанные структуры tracr-РНК, образующие пары оснований с этими повторами. Tracr-РНК для Alicyclobacillus acidoterrestric был идентифицирован с помощью секвенирования РНК. Для остальных локусов предполагаемые tracr-РНК идентифицированы на основе присутствия последовательности антипрямого повтора (DR) (Anti-DR). Последовательности антипрямых повторов были идентифицированы с помощью Geneious (www.geneious.com) путем поиска последовательностей внутри каждого соответствующего локуса CRISPR с высокой гомологией к DR. 5'- и 3'-концы каждой предполагаемой tracr-РНК были определены с помощью вычислительного прогнозирования сайтов старта бактериальной транскрипции и терминации с помощью соответственно BPROM (www.softberry.com) и ARNOLD (rna.ig-mors.u-psud.fr/toolbox/amold/). Предсказания кофолдинга получены с помощью Geneious, 5'-концы выделены синим, 3'-концы выделены оранжевым.
[131] Фиг. 42A-42N. Рисунок 42A-B содержит таблицу, демонстрирующую результаты анализа локусов типа VI (кодирующих белок C2c2). *CRISPR - CRISPR повторы; unk - гипотетический белок; > - направление кодирующей последовательности гена; [D] - вырожденный повтор (определены, где возможно); [T] - tracr-РНК, фиг. 42C-I демонстрируют результаты анализа CRISPR-последовательностей локусов типа VI (кодирующих белок C2c2) как описано в настоящем описании (раздел CRISPR является частью базового результата работы pilercr (см. описание результата работы на сайте pilercr: http://www.drive5.com/pilercr/); фолдинг повторов выполнен с помощью mfold (см. описание результата работы на сайте mfold: http://mfold.rna.albany.edu/?q=mfQld/DNA-Folding-Form); результаты фолдинга повторов и анализа CRISPR-последовательностей помещены после детального описания каждого случая; см. расположение CRISPR по ссылке в таблице на фиг. 42 A-В. Фиг. 42J иллюстрирует классификацию CRISPRmap CRISPR повторов локусов типа VI (кодирующих белок C2c2) с помощью, как описано в настоящем описании CRISPRmap (детали см. http://rna.informatik.uni-freiburg.de/CRISPRmap/Input.jsp). Фиг. 42K-L иллюстрирует вырожденные повторы локусов типа VI (кодирующие белок C2c2) как описано в настоящем описании, обнаруженные с помощью алгоритма по поиску CRISPR-последовательностей (http://crispr.u-psud.fr/Server/). Столбец нормальных повторов содержит нормальный повтор, столбец спейсеров - последний спейсер, столбец downstream - область низлежащих последовательностей, начиная с вырожденного повтора (250 п.н.); номер последовательности соответствует числовому значению CRISPR-последовательности в соответствующем локусе (см. таблицу на фиг. 42 A-В); выделенная желтым область имеет полное совпадение между нормальным повтором и вырожденным повтором (при несовпадении с другой частью вырожденного повтора). Фиг. 42M-N иллюстрирует предсказанные структуры tracr-РНК, образующие пары оснований с этими повторами. Предполагаемые tracr-РНК были идентифицированы на основе присутствия последовательности антипрямого повтора (DR) (Anti-DR). Последовательности антипрямых повторов были идентифицированы с помощью Geneious (www.geneious.com) путем поиска последовательностей внутри каждого соответствующего локуса CRISPR с высокой гомологией с DR. 5'- и 3'-концы каждой предполагаемой tracr-РНК были определены с помощью вычислительного прогнозирования сайтов старта бактериальной транскрипции и терминации с помощью BPROM (www.softberry.com) и ARNOLD (rna.ig-mors.u-psud.fr/toolbox/amold/) соответственно. Предсказания кофолдинга получены с помощью Geneious, 5'-концы выделены синим, 3'-концы выделены оранжевым.
[132] Фиг. 43A-43F. Рисунок 43 A-B содержит таблицу, демонстрирующую результаты анализа локусов типа V-С (кодирующих белок C2c3). *CRISPR - CRISPR-повторы; unk - гипотетический белок; > - направление кодирующей последовательности гена; [D] - вырожденный повтор (определены, где возможно). Фиг. 43C - демонстрируют результаты анализа CRISPR-последовательностей локусов типа V-С (кодирующих белок C2c3) как описано в настоящем описании (раздел CRISPR является частью базового результата работы pilercr (см. описание результата работы на сайте pilercr: http://www.drive5.com/pilercr/); фолдинг повторов выполнен с помощью mfold (см. описание результата работы на сайте mfold: http://mfold.ma.albany.edu/?q=mfQld/DNA-Folding-Form); результаты фолдинга повторов и анализа CRISPR-последовательностей помещены после детального описания каждого случая; см. расположение CRISPR по ссылке в таблице на фиг. 43 A-В. Показаны статистически значимые совпадения BLAST для спейсера с прокариотами и их вирусами. Фиг. 43E иллюстрирует классификацию CRISPRmap CRISPR повторов локусов типа V-С (кодирующих белок C2c3) как описано в настоящем описании с помощью CRISPRmap (детали см. http://rna.informatik.uni-freiburg.de/CRISPRmap/Input.jsp). Фиг. 43F иллюстрирует вырожденные повторы локусов типа V-С (кодирующие белок C2c3) как описано в настоящем описании, обнаруженные с помощью алгоритма по поиску CRISPR-последовательностей (http://crispr.u-psud.fr/Server/). Столбец нормальных повторов содержит нормальный повтор, столбец спейсера - последний спейсер, столбец downstream - область низлежащих последовательностей, начиная с вырожденного повтора (250 п.н.); номер последовательности соответствует номеру CRISPR-последовательности в соответствующем локусе (см. таблицу на фиг. 43A-В); выделенная желтым область имеет полное совпадение между нормальным повтором и вырожденным повтором (при несовпадении с другой частью вырожденного повтора).
[133] На фиг. 44A-44E приведен полный список локусов CRISPR-Cas в геномах, где были обнаружены белки C2c1 или C2c2. Гены белков C2c1 и C2c2 выделены желтым.
[134] На фиг. 45A-45C проиллюстрировано выравнивание локусов Listeria, кодирующих предполагаемую систему CRISPR-Cas типа VI. Выровненный синтетический участок, соответствующий контигу AODJ01000004.1 Listeria weihenstephanensis FSL R9-0317 с координатами 42281-46274 и контигу JNFB01000012.1 Listeria newyorkensis, штамм FSL M6-0635, с координатами 169489-173541. Обозначения: ген C2c2 выделен голубым, повторы CRISPR - пурпурным, вырожденные повторы - пурпурным, спейсеры - жирным.
[135] На фиг. 46 проиллюстрировано два локуса C2c2 Carnobacterium gallinarum.
[136] На фиг. 47 схематически изображен уровень экспрессии для двух CRISPR-последовательностей в направлении гена C2c2 в первом локусе C2c2.
[137] На фиг. 48 схематически изображен уровень экспрессии для CRISPR-последовательностей с направлением транскрипции в сторону гена C2c2 во втором локусе C2c2.
[138] Фиг. 49A-49B. Фиг. 49A-49B иллюстрируют экспрессию и процессинг локусов C2c2. Фиг. 49A: секвенирование РНК локуса C2c2 Listeriaseeligeria, серовар 1/2b, штамм SLCC3954, экспрессированного в E.coli. Данный локус имеет высокий уровень экспрессии, процессированная cr-РНК содержит прямой повтор на 5'-конце длиной 29 п.н. и спейсер длиной 15-18 п.н. Для предполагаемой tracr-РНК экспрессия не установлена. In silico фолдинг РНК прямого повтора процессированной cr-РНК продемонстрировал стабильную шпилечную структуру. Фиг. 49B: нозерн-блоттинг РНК локуса C2c2 Leptotrichia shahii DSM 19757, экспрессированного в E.coli, демонстрирует процессированные cr-РНК с прямыми повторами на 5'-конце. Стрелки обозначают положения зондов и их направление.
[139] Фиг. 50A-50C. Фиг. 50A-C иллюстрируют экспрессию и процессинг локуса C2c2 Leptotrichia shahii DSM 19757. Фиг. 50A: секвенирование РНК локуса Leptotrichia shahii DSM 19757, экспрессированного в E.coli демонстрирует процессинг CRISPR-последовательности в направлении от 3'-конца к 5'-концу (направление этого локуса). cr-РНК процессируются таким образом, что имеют 5'-концевой прямой повтор длиной 28 п.н. и спейсеры длиной 14-28 п.н. Фиг. 50B: секвенирование РНК эндогенного локуса C2c2 Leptotrichia shahii DSM 19757 демонстрирует близкие результаты с изображенными на фиг. 50A. Фиг. 50C: In silico фолдинг прямого повтора (DR) в cr-РНК L. shahii предсказывает стабильную вторичную структуру.
[140] На фиг. 51 проиллюстрирован эволюционный сценарий для систем CRISPR-Cas. Предполагается, что белок Cas8 возник путем инактивации Cas10 (обозначен белым X), что сопровождалось значительным ускорением эволюции. Сокращения: TR, концевые повторы; TS, концевые последовательности, HD, семейство эндонуклеаз HD; HNH, семейство эндонуклеаз HNH, RuvC, семейство эндонуклеаз RuvC; HEPN, предполагаемая эндорибонуклеаза суперсемейства HEPN. Показанные серым области генов и белков обозначают последовательности, которые были закодированы в соответствующих мобильных элементах, но были утрачены в ходе эволюции систем CRISPR-Cas.
[141] На фиг. 52 изображена организация локуса гена C2c2, включая домены, принадлежащие к суперсемейству HEPN. Большая часть доменов HEPN содержит консервативные мотивы и представляет собой металл-независимые эндоРНК-азы.
[142] На фиг. 53 изображен результат анализа секвенирования РНК для эндогенного локуса из Leptotrichia shahii DSM 19757.
[143] На фиг. 54 схематически представлена схема эксперимента in vivo с использованием E.coli, экспрессирующей LshC2c2, с целью идентификации мотивов обедненными последовательностями. Библиотека PAM, обеспечивающая устойчивость к ампициллину, была перенесена в E.coli. Плазмиды, несущие мотивы последовательностей, содержащие детерминантную последовательность PAM, утрачены и, следовательно, не способны обеспечивать устойчивость к ампициллину. Мотивы последовательности PAM идентифицированы исходя из отсутствия определенных последовательностей.
[144] Фиг. 55 Идентификация последовательности PAM. Последовательности, для которых наблюдается обеднение, позволяют определить нуклеотиды 5'-PAM.
[145] На фиг. 56 изображено нацеливание на эндогенную мишень в E.coli. Интерференция установлена по уменьшению числа колониеобразующих единиц (КОЕ) на 20 нг плазмид. Интерференция наблюдалась в E.coli, содержащих LshC2c2, но не контрольной плазмиде pACYC184. Усиление интерференции связано с транскрипцией целевой последовательности.
[146] На фиг. 57 проиллюстрирована очистка C2c2 с использованием способа очистки с His-меткой с последующими одним этапом (слева) или тремя этапами (справа) гель-фильтрации, а также одна полоса красителя кумасси, соответствующая 168 кДа.
[147] На фиг. 58 изображены компоненты экспериментов in vitro с очищенным Lsh C2c2 и очищенной с помощью FPLC РНК-мишенью. Компонент "166" обозначает некомплементарную последовательность-мишень. "E" обозначает EDTA. Расщепление cr-РНК наблюдается в случае присутствия C2c2.
[148] На фиг. 59 изображены эксперименты in vitro с очищенным C2c2, РНК-мишенью и cr-РНК, имеющими спейсеры длиной 12-26 нуклеотидов. Наблюдается расщепление cr-РНК, имеющих спейсеры длиной 28 или 24 п.н.
[149] На фиг. 60 изображен анализ сдвига электрофоретической подвижности (EMSA), позволяющий выявить комплексы состоящие из белков и нуклеиновых кислот.
[150] На фиг. 61 изображено нацеливание in vivo на транскрибированный красный флуоресцентный белок (RFP), с использованием спейсеров RFP, соответствующих или комплементарных РНК. Спейсеры, нацеливающие на транскрибируемый RFP, были клонированы в локус LshC2c2 с последующей экспрессией в E.coli, несущих плазмиду, экспрессирующую RFP, или контрольную плазмиду pUC19. Интерференция была определена на основе колониеобразующих единиц (КОЕ) на 20 п.н. трансформированной плазмиды.
[151] На фиг. 62 изображает зависимую от цепи интерференцию с плазмидой, несущей RFP-мишень. Интерференция была измерена как число колониеобразующих единиц (КОЕ) для 6 RFP-мишеней (слева). Наблюдалась зависимость интерференции от цепи, на которую было осуществлено нацеливание, и присутствия нуклеотида PAM. Интерференция с ненацеленной контрольной плазмидой pUC19 не наблюдалась (справа).
[152] Фиг. 63A-63C. На фиг. 63 изображен эффект белка C2c2 с нацеливающей РНК, комплементарной или некомплементарной экспрессированной РНК, на экспрессию RFP. А - схема нацеливания на RFP в гетерологической системе E.coli. Локусы LshC2c2, несущие спейсеры для нацеливания на RFP в различных последовательностях PAM, были встроены в экспрессирующую RFP E.coli. B - количественная оценка нацеливания на RFP в E.coli для множественных спейсеров для нацеливания A-, C- или U PAM. Экспрессия RFP была измерена с помощью поточной цитометрии. C - количественная оценка нацеливания на RFP в E.coli. Были встроены спейсеры с различными последовательностями PAM для нацеливания на некодирующую цепь ("ДНК") или кодирующую цепь ("РНК") гена RFP, и экспрессия RFP была измерена с помощью поточной цитометрии.
[153] На фиг. 64 изображен процессинг последовательностей прямых повторов (DR) при помощи LshC2c2. LshC2c2 процессирует прямые повторы (DR) на 5'-конце.
[154] На фиг. 65 изображена стратегия исследования выбора мишени. Мишень 1 (T1) содержит G-PAM, мишень 3 (T3) содержит C-PAM.
[155] На фиг. 66 изображены результаты реакции расщепления РНК белком C2c2, нацеленным на T3 (см. Фиг. 65). Компоненты реакции обозначены.
[156] На фиг. 67 представлены результаты реакции расщепления РНК белком C2c2, нацеленным на T1 (см. Фиг. 65). Компоненты реакции обозначены.
[157] На фиг. 68 изображено C2c2-опосредованное расщепление, нацеленное на РНК, экспрессированную с матрицы ДНК in vitro. Компоненты реакции обозначены. Дорожки 2-8: C2c2-опосредованное расщепление нацелено на T1 (см. Фиг. 65), дорожки 9-15: C2c2-опосредованное расщепление нацелено на последовательность, обратно комплементарную T1.
[158] На фиг. 69 изображено C2c2-опосредованное расщепление, нацеленное на РНК, экспрессированную с матрицы ДНК in vitro. Компоненты реакции обозначены. Дорожки 2-8: C2c2-опосредованное расщепление нацелено на T3 (см. Фиг. 65).
[159] На фиг. 70 изображены установленные размеры фрагмента РНК, для которого наблюдается C2c2-опосредованное расщепление, нацеленное на T1 или T3 (см. Фиг. 65).
[160] На фиг. 71 изображено C2c2-опосредованное расщепление РНК-мишеней T1 и T3. Наблюдаются множественные продукты расщепления и значительное сокращение интенсивности полосы мишени. Буфер 1: 40 мМ Tris-HCl (pH 7,9), 6 мМ MgCL2, 83 мМ NaCl, 1 мМ DTT, rNTP, полимераза T7. Буфер 2: 25 мМ Tris-HCl (pH 7,5), 10 мМ MgCL2, 83 мМ NaCl, 5 мМ DTT. Буфер 2 воспроизводит условия реакции без ДНК-матрицы.
[161] На фиг. 72 показано, что мутации в любом домене HEPN препятствуют нацеливанию на РНК.
[162] На фиг. 73 представлен схематический обзор скрининга PAM РНК с использованием интерференции фага MS2. Библиотека, состоящая из спейсеров, нацеливающих на все возможные последовательности в РНК-геноме фага MS2, была клонирована в CRISPR-последовательность LshC2c2. Популяция клеток с этой библиотекой была обработана фагом и помещена в культуру для дальнейшего отбора выживших клеток. Частоту спейсеров сравнили с контрольной группой, не обработанной фагом, и обогащенные фагом спейсеры использовали для создания логотипов последовательностей.
[163] На фиг. 74 показано, что скрининг интерференции РНК-фага демонстрирует как значительное обогащение, так и обеднение спейсеров LshC2c2 PAM РНК, полученными с использованием интерференции фага MS2.
[164] Фиг. 75A-75J. На фиг. 75A-C представлена идентификация правой последовательности PAM, состоящей из единственного основания, для LshC2c2 с помощью скрининга PAM РНК с использованием интерференции фага MS2. Более конкретно, показано наличие справа H-PAM (не G). На фиг. 75C представлена количественная оценка анализа бляшек фага MS2, подтверждающего наличие PAM. Множественные спейсеры, направленные на каждый PAM, были клонированы в локус LshC2c2. Разведения фага были нанесены на чашки Петри с бактериями, и интерференция была определена количественно по максимальному разведению, при котором не наблюдалось литических бляшек. Фиг. 75D демонстрирует репрезентативные изображения, полученные при подсчете литических бляшек фага MS2, показывающие уменьшение формирования бляшек при наличии Н-PAM в спейсерах, но не при наличии G-PAM в спейсерах. На фиг. 75E представлено присутствие нуклеазной активности при наличии G-PAM в спейсерах и устойчивости при наличии H-PAM в спейсерах. Фиг. 75F иллюстрирует схему РНК-мишени, показывая участок протоспейсера и соответствующую cr-РНК. На фиг. 75G в денатурирующем геле показана ферментативная активность LshC2c2 по расщеплению оцРНК с использованием в качестве мишени РНК, меченных на 5'-конце IRDye 800 и на 3'-конце - Cy5. Наблюдаются четыре независимых участка расщепления. На фиг. 75 H в денатурирующем геле показан Н-РАМ (не G). Ферментативная активность по расщеплению оцРНК зависит от нуклеотида непосредственно на 3'-конце мишени. PAM был протестирован путем внесения мутаций в прилежащий нуклеотид и использования той же самой cr-РНК/мишени. На фиг. 75I изображена схема, показывающая расположенные по типу черепицы cr-РНК, чтобы продемонстрировать повторное нацеливание (перенацеливание) LshC2c2 и H-PAM (вверху) и соответствующий денатурирующий гель (внизу). Пять различных cr-РНК были протестированы для каждого возможного нуклеотида. В денатурирующем геле также продемонстрировано, что H-PAM использует различные CRISPR-РНК, расположенные по типу черепицы вдоль оцРНК-мишени. Пять различных cr-РНК были протестированы для каждого возможного нуклеотида. На фиг. 75J в денатурирующем геле показано тестирование на присутствие мотива спейсера. Были проверены три cr-РНК с каждым возможным последним нуклеотидом в качестве последнего основания в последовательности спейсера.
[165] На фиг. 76 продемонстрирована рестрикция РНК фага MS2, посредством LshC2c2. Клонированные спейсеры, нацеливающие на каждый из четырех генов фага MS2, с С- и G- PAM. G-PAM требуют более высоких концентраций фага для развития литических бляшек.
[166] На фиг. 77 продемонстрировано, что нацеливание на транскрипты RFP у бактерий замедляет скорость роста.
[167] Фиг. 78A-78D. На фиг. 78 A-В продемонстрировано, что LshC2c2 эффективно расщепляет РНК. Получение данных с использованием Minigel; не флюоресценция. На фиг. 78C продемонстрировано, что расщепление оцРНК LshC2c2 с использованием 5'-маркированных и 3'-маркированных мишеней было проанализировано в обозначенные моменты времени. На фиг. 78D представлена количественная оценка данных фиг. 78B.
[168] Фиг. 79: белок LshC2c2 и cr-РНК были инкубированы и последовательно разведены. Расщепление оцРНК было оценено с использованием обозначенных концентраций комплекса.
[169] На фиг. 80 продемонстрировано, что LshC2c2 эффективно расщепляет РНК. Считывание в 20-см слое геля; флуоресцентное изображение на 700 нм. Расщепление наблюдается только с меньшей РНК-мишенью длиной 85 нуклеотидов вместо обычной мишени длиной 173 нуклеотидов.
[170] На фиг. 81 представлено картирование фрагментов расщепления.
[171] На фиг. 82 представлено секвенирование РНК нуклеазной реакции in vitro.
[172] На фиг. 83 показано, что LshC2c2 не расщепляет нетранскрибированную или транскрибированную ДНК в RNAP E.coli при анализе in vitro. Анализ организован согласно Samai et al. (Cell, 2015). Использована мишень длиной 200 п.н. (соответствующая РНК-мишени фиг. 82). Инкубация в течение 1 ч при 37°C необходима для параллельной транскрипции и расщепления после формирования открытого комплекса.
[173] На фиг. 84 показано, что LshC2c2 не расщепляет оцДНК in vitro. Инкубация в течение 1 ч при 37°C, оцДНК-версия t3 G-PAM и последовательность, обратно комплементарная (RC) ей.
[174] Фиг. 85A-85B. На фиг. 85 представлено, что C2c2 не расщепляет дцДНК (A) и оцДНК (B). Гель для полос был экстрагирован и подготовлен к секвенированию последнего поколения с помощью Illumina MiSeq.
[175] На фиг. 86 показано, что LshC2c2 имеет 3'-G-PAM для расщепления РНК. Та же самая мишень с варьирующими PAM. (Показанная последовательность PAM является обратно комплементарной, так, что C на 5'-конце соответствует G на 3'-конце).
[176] На фиг. 87 показано, что LshC2c2 не требует малой РНК для расщепления РНК.
[177] На фиг. 88 показано, что LshC2c2 является перепрограммируемым и чувствительным к PAM.
[178] На фиг. 89 показано, что LshC2c2 является перепрограммируемым и чувствительным к PAM.
[179] Фиг. 90A-90B. На фиг. 90А и В показано, что расщепление оцРНК было проанализировано с помощью cr-РНК с различной длиной спейсера.
[180] Фиг. 91A-91C. На фиг. 91 А, B и C показано, что расщепление оцРНК, было проанализировано с использованием cr-РНК с различной длиной прямых повторов (DR). Расщепление РНК зависит от длины cr-РНК (фиг. 91 А, B). Расщепление РНК зависит от длины прямых повторов (DR) (фиг. 91C-E).
[181] Фиг. 92A-92D. На фиг. 92А, В, C и D представлены модификации стеблевой структуры CRISPR LshC2c2 и показано, что стеблевая структура подвержена отдельным заменам оснований, но активность нарушается только с изменением большей части вторичной структуры. Эксперименты по укорочению прямых повторов (DR) также указывают, что нарушение стеблевой структуры прекращает расщепление.
[182] Фиг. 93A-93D. На фиг. 93 А, В, C и D представлены модификации шпилечной структуры CRISPR LshC2c2 и показано, что шпилечная структура cr-РНК подвержена заменам оснований и удлинению, но не укорочению.
[183] На фиг. 94 показано, что C2c2 процессирует свою собственную последовательность. Буфер 1: 40 мМ Tris-HCl (pH 7,9), 6 мМ MgCl2, 70 мМ NaCl, 1 мМ DTT. Буфер 2: 40 мМ Tris-HCl (pH 7,3), 6 мМ MgCl2, 70 мМ NaCl, 1 мМ DTT, ингибитор РНК-азы мыши. LshC2c2 расщепляет как последовательность 1, так и последовательность 2 (последовательности различной длины) в обоих протестированных буферах. Расщепление становится очевидным по появлению более низких полос.
[184] На фиг. 95 показано, что мутанты по домену HEFN продолжают обладать способностью процессировать CRISPR-последовательность C2c2.
[185] На фиг. 96 показано, что процессинг последовательности C2c2 в E.coli требует присутствия белка C2c2.
[186] Фиг. 97A-97D. На фиг. 97A представлена схема, которая показывает различные мутации в домене HEPN в белке C2c2. Схема локуса и белка LshC2c2 показывает консервативные остатки в доменах HEPN. Фиг. 97В и C показывают, что каждый из мутантов по домену HEPN демонстрирует значительное снижение активности. C, вверху: в денатурирующем геле показаны консервативные остатки мотива HEPN, необходимые для расщепления оцРНК. C, внизу: количественная оценка анализа литических бляшек фага MS2 с мутантами в каталитических остатках домена HEPN. Локусы с мутантным LshC2c2 были неспособны защитить от фага. D, количественная оценка нацеливания на REP в E.coli с мутантами по аргинину в каталитических остатках домена HEPN. Локусы с мутантным LshC2c2 были неспособны к нокдауну RFP.
[187] На фиг. 98 представлен "эффект свидетеля". После активации C2c2 становится активным и деградирует другие РНК в образце. Верхний ряд: L=длинная мишень, small=короткая мишень, LC=длинная мишень с С-PAM. Нижний ряд: эффект присутствия или отсутствия активирующей мишени на расщепление различных мишеней-"свидетелей".
[188] Фиг. 99A-C. На фиг. 99А показано, что белок C2c2, мутантный по домену HEPN, продолжает обладать способностью связывать cr-РНК. На фиг. В и C показано влияние вторичной структуры РНК на продукт расщепления РНК-мишени.
[189] Фиг. 100A-100B. На фиг. 100A показывает эффект различных двухвалентных катионов на активность C2c2. Фиг. 100В показывает влияние наличия или отсутствия магния на титрование cr-РНК.
[190] На фиг. 101 продемонстрирована активность расщепления оцРНК белком LshC2c2 в денатурирующем геле с использованием мишени РНК, меченной на 5'-конце IRDye 800 и на 3'-конце - Cy5. Наблюдаются четыре независимых участка расщепления. Из этой фиг. также видно, что хелатирование ионов Mg2+ влияет на расщепление оцРНК под действием белка С2с2.
[191] На фиг. 102 показано, что C2c2 отщепляет 3'-конец мишени.
[192] На фиг. 103 показано, что C2c2 отщепляет 3'-конец мишени.
[193] На фиг. 104 показано перепрограммирование C2c2 с помощью cr-РНК.
[194] На фиг. 105 показано перепрограммирование C2c2 с помощью cr-РНК.
[195] На фиг. 106 показано перепрограммирование C2c2 с помощью cr-РНК.
[196] На фиг. 107 представлена IVC (нуклеазная реакция in vitro) длинной мишени.
[197] На фиг. 108 представлен "эффект свидетеля". В отсутствие магния наблюдается уменьшение расщепления.
[198] На фиг. 109 проиллюстрировано выравнивание последовательностей следующих ортологов: DSM 19757 C2c2Leptotrichia shahii; SB 1003 (RcS) Rhodobacter capsulatus; R121 (RcR) Rhodobacter capsulatus; DE442 (RcD) Rhodobacter capsulatus; MA2020 (Lb(X)) бактерий Lachnospiraceae; NK4A179 (Lb(X) бактерий Lachnospiraceae; DSM 10710 (CaC) [Clostridium] aminophilum; NK4A144 (Lb(X) бактерий Lachnospiraceae; F0279 (Lew) Leptotrichia wadei, F0279 (Lew7) Leptotrichia wadei; DSM 4847 (Cg) Carnobacterium gallinarum; WB4 (Pp) Paludibacter propionicigenes; 2b (Ls) Listeria seeligeri серовара 1; FSL R9-0317 (Liw) Listeria weihenstephanensis; и FSL М6-0635 (Lib) бактерий Listeria.
[199] На фиг. 110 представлены консервативные домены HEPN белков C2c2.
[200] На фиг. 111 показано, что мутанты С2с2 по домену HEPN сохраняют целевую активность связывания. Верхний ряд: использование способа сдвига электрофоретической подвижности для комплекса LshC2c2-cr-РНК дикого типа в отношении являющейся мишенью оцРНК и не являющейся мишенью комплементарной оцРНК. Нижние ряды: использование способа сдвига электрофоретической подвижности для комплекса LshC2c2-cr-РНК мутанта по домену HEPN R1278A в отношении являющейся мишенью оцРНК и не являющейся мишенью комплементарной оцРНК.
[201] Фиг. 112A-112D. На фиг. 112 представлен эффект нарушения комплементарности РНК-мишени и cr-РНК на РНК-азную активность LshC2c2. A. Количественная оценка одиночных нарушений комплементарности в различных положениях спейсера с использованием способа анализа литических бляшек фага MS2. Отдельные нарушения комплементарности имеют минимальный эффект на интерференцию фага. Положения нарушений комплементарности показаны красным; все нарушения комплементарности вызваны трансверсиями. B. Количественная оценка двойных нарушений комплементарности в различных положениях спейсера с использованием способа анализа литических бляшек фага MS2. Последовательные двойные нарушения комплементарности в середине спейсера прекращают интерференцию фага. Положения нарушений комплементарности показаны красным; все нарушения комплементарности вызваны трансверсиями. C. Расщепление in vitro белком Lshc2c2 для анализа одиночных нарушений комплементарности в cr-РНК. Одиночные нарушения комплементарности имеют минимальный эффект на расщепление оцРНК. Положения нарушений комплементарности показаны красным; все нарушения комплементарности вызваны трансверсиями. D. Расщепление in vitro белком Lshc2c2 для анализа двойных нарушений комплементарности в cr-РНК. Последовательные двойные нарушения комплементарности в середине спейсера прекращают активность LshC2c2. Положения нарушений комплементарности показаны красным; все нарушения комплементарности вызваны трансверсиями.
[202] Фиг. 113. Расщепление трех оцРНК-мишеней, имеющих одну и ту же последовательность, являющуюся мишенью cr-РНК, фланкированную различными последовательностями. Показана вторичная структура, и последовательность каждой из трех мишеней (вверху), паттерны расщепления белком C2c2 для каждой мишени продемонстрированы с использованием 10% геля PAGE (внизу).
[203] Фиг. 114A-114B. Картирование сайтов расщепления C2c2 с помощью секвенирования РНК: A, по положению; В, по вторичной структуре. A Графики для частоты наблюдения концов расщепления согласно данным секвенирования РНК для 5'-фиксированных фрагментов. Данные соотнесены со вторичной структурой (В). В, Сайты расщепления мишеней 1 и 3 картированы с помощью секвенирования РНК в реакции расщепления. Частота наличия концов каждого фрагмента (фиксированного на 5'-конце) соотнесена со значениями z-оценки и спроецирована на вторичную структуру мишени.
[204] На фиг. 115A-115I представлены вторичные структуры окружения мишени, которые в дальнейшем могут быть использованы для оценки эффективности расщепления C2c2 и картирования сайтов расщепления.
[205] Фиг. 116A-116E. Гетерологическая экспрессия локуса C2c2 Leptotrichia shahii опосредует значительную интерференцию РНК фага в Escherichia coli. (A) Схема скрининга интерференции бактериофага MS2. Библиотека, содержащая спейсеры, нацеливающие на все возможные последовательности в РНК-геноме фага MS2, была клонирована в матрицу CRISPR LshC2c2. Клетки, трансформированные нацеливающей на MS2 библиотекой спейсеров, были далее обработаны раствором с фагом и помещены в культуру для последующего отбора выживших клеток. Частоту встречаемости спейсеров сравнили с таковой для контрольных клеток (необработанных фагом), и спейсеры, для которых наблюдалось обогащение после обработки фагом, были использованы для создания логотипа PAM-последовательности. (B) График типа "ящик с усами", иллюстрирующий распределение нормализованных частот встречаемости cr-РНК при условии обработки фагом и контроля в повторности (n=2). Границами ящика являются первый и третий квартили, концы усов соответствуют 1 и 99 перцентилям. Значение среднего обозначено красной горизонтальной чертой. (C) Логотип, созданный на основе последовательностей, фланкирующих 3'-конец протоспейсеров, соответствующих тем спейсерам, для которых наблюдается обогащение, выявляющее присутствие 3'-концевой H-PAM (не G-PAM). (D) Анализ литических бляшек был использован для подтверждения функциональной значимости интерференции H-PAM в MS2. Все протоспейсеры, фланкированные PAM, отличной от П-PAM, демонстрируют выраженную интерференцию в фаге. Спейсеры были разработаны для нацеливания на ген mat фага MS2, их последовательности приведены ниже изображений литических бляшек; использованный спейсер в ненацеливающем контроле не комплементарен ни одной последовательности в геномах E.coli или MS2. Литические бляшки были получены в ходе серии полулогарифмических разведений. (E) Количественный анализ литических бляшек фага MS2, подтверждающих необходимость в H-PAM (не G). Для каждого PAM 4 было разработано нацеливающих на MS2 спейсера. Разведения фага были посеяны на бактериальный газон в серии полулогарифмических разведений, интерференция была оценена на основе максимального разведения, при котором не происходило образование литических бляшек. Каждая точка диаграммы рассеяния представляет собой среднее для трем биологических повторений и соответствует единственному спейсеру. Линии соответствуют среднему для 4 спейсеров для каждого PAM, ошибки показаны в виде s.e.m.
[206] Фиг. 117A-117D. LshC2c2 и cr-РНК опосредуют РНК-направляемое расщепление оцРНК. (A) Схема оцРНК-субстрата, на который нацелена cr-РНК, область протоспейсера выделена голубым, и PAM обозначен лиловой полосой. (B) В денатурирующем геле показано опосредованное cr-РНК расщепление оцРНК белком LshC2c2. Мишень оцРНК является меченной на 5'-конце IRDye 800 или на 3'-конце Cy5. Расщепление требует присутствия cr-РНК и прекращается при добавлении ЭДТА. Наблюдаются четыре участка расщепления. (С) В денатурирующем геле показана необходимость H-PAM (не G). Четыре идентичных, за исключением основания в PAM (обозначено лиловым X в схеме), оцРНК-субстрата использованы для реакции расщепления in vitro, активность по расщеплению оцРНК зависит от нуклеотида непосредственно на 3'-конце мишени. (D) Схема, показывающая пять протоспейсеров для каждого PAM на оцРНК-мишени (вверху). В денатурирующем геле показана направляемая cr-РНК активность белка LshC2c2 по расщеплению оцРНК. cr-РНК соответствуют нумерации протоспейсеров.
[207] Фиг. 118A-118I. Участки расщепления C2c2 определены вторичной структурой и последовательностью РНК-мишени (A) Схема мишеней гомополимерной оцРНК. Протоспейсер обозначен светло-голубой полосой. Участки гомополимера, образованные основаниями A (зеленым) и U (красным), перемежаются отдельными основаниями G (оранжевым) и C (фиолетовым). (B) В денатурирующем геле показаны паттерны опосредованного C2c2-cr-РНК расщепления каждого гомополимера. (C) В денатурирующем геле показаны паттерны опосредованного C2c2-cr-РНК расщепления трех негомополимерных мишеней оцРНК (1, 4, 5), которые имеют одинаковый протоспейсер, но фланкированы различными последовательностями. Вне зависимости от идентичных протоспейсеров, различные фланкирующие последовательности привели к различным паттернам расщепления (D, F и H). Участки расщепления негомополимерных мишеней оцРНК 1 (D), 4 (F) и 5 (H) были картированы с использованием секвенирования РНК продуктов расщепления. Частота расщепления по каждому основанию окрашена согласно z-оценке и показана на предсказанной вторичной структуре кофолдинга оцРНК и cr-РНК. Фрагменты, используемые для проведения анализа частот встречаемости, содержат полные 5'-концы. 5'- и 3'-концы мишени оцРНК обозначены соответственно голубыми и красными контурами. 5'- и 3'-концы спейсера (желтым контуром) выделены голубым и оранжевым соответственно. (E, G и I) Графики частот встречаемости участков расщепления для каждого положения оцРНК-мишеней 1, 4 и 5 для всех прочтений, начинающихся на 5'-конце. Область протоспейсера выделена голубым.
[208] Фиг. 119A-119E. Два домена HEPN C2c2 необходимы для направляемого CRISPR-РНК расщепления оцРНК, но не для направляемого CRISPR-РНК связывания оцРНК. (A) Схема локуса LshC2c2 и организация доменов белка LshC2c2, показывающая консервативные остатки в доменах HEPN. (B) Определение числа литических бляшек фага MS2 было выполнено с использованием мутантов по каталитическим остаткам HEPN. Для каждого мутанта был использован один и тот же нацеливающий cr-РНК протоспейсер 35. (C) В денатурирующем геле показаны консервативные остатки мотива HEPN, необходимые для направляемого cr-РНК расщепления оцРНК. (D) Результаты способа сдвига электрофоретической подвижности (EMSA), использованного для оценки сродства комплекса LshC2c2-CRISPR-РНК дикого типа в отношении являющейся мишенью (слева) и не являющейся мишенью (справа) оцРНК-субстрата. Не являющаяся мишенью оцРНК-субстрат обратно комплементарна являющейся мишенью оцРНК. ЭДТА добавлена в реакционную среду с целью неспецифически снизить расщепление. (E) Результаты использования способа сдвига электрофоретической подвижности для комплекса LshC2c2(R1278A)-cr-РНК в сравнении с являющейся мишенью оцРНК и не являющейся мишенью комплементарной оцРНК (последовательности субстрата те же, что в D).
[209] Фиг. 120A-120B. Направляемая РНК РНК-азная активность белка LshC2c2 зависит от спейсера и длины прямых повторов. (A) В денатурирующем геле показана функция направляемого cr-РНК расщепления оцРНК 1 в зависимости от длины спейсера. (B) В денатурирующем геле показана функция направляемого cr-РНК расщепления оцРНК в зависимости от длины повтора.
[210] Фиг. 121A-121B. Направляемая РНК РНК-азная активность белка LshC2c2 зависит от структуры прямых повторов и аминокислотной последовательности. (A) Схема, изображающая модификации стеблевой структуры прямого повтора cr-РНК (вверху). Измененные основания выделены красным. В денатурирующем геле показано направляемое cr-РНК расщепление оцРНК 1 посредством каждой модифицированной cr-РНК (внизу). (B) Схема, изображающая модификации шпилечной структуры прямого повтора cr-РНК (вверху). Измененные основания обозначены красным, длина удаленных последовательность показана стрелками. В денатурирующем геле показано направляемое cr-РНК расщепление оцРНК посредством каждой из модифицированных cr-РНК (внизу).
[211] Фиг. 122A-122D. Влияние нарушений комплементарности РНК-мишени и cr-РНК на РНК-азную активность белка LshC2c2. (A) Подсчет числа литических бляшек фага MS2 для изучения единичных нарушений комплементарности в различных положениях спейсера. Единичные нарушения комплементарности имеют минимальный эффект на интерференцию фага. Положение и идентичность нарушений комплементарности обозначены красным. (B) Подсчет числа литических бляшек фага MS2 для изучения двойных нарушений комплементарности в различных положениях спейсера. Последовательные двойные нарушения комплементарности в середине спейсера прекращают интерференцию фага. Положение и идентичность нарушений комплементарности обозначены красным. (C) Схема, показывающая положение и идентичность нарушений комплементарности (красным) в спейсере cr-РНК (вверху). В денатурирующем геле показано расщепление оцРНК 1, направляемое cr-РНК с единичными нарушениями комплементарности в спейсере (внизу). (D) Схема, показывающая положение и идентичность двойных нарушений комплементарности (красным) в спейсере cr-РНК (вверху). В денатурирующем геле показано расщепление оцРНК 1, направляемое cr-РНК с парами нарушений комплементарности в спейсере (внизу).
[212] Фиг. 123A-123E. Нокдаун мРНК RFP путем перенацеливания LshC2c2. (A) Схема, показывающая направляемый cr-РНК нокаут RFP в E.coli, осуществляющей гетерологическую экспрессию локуса LshC2c2. Три нацеливающих на RFP спейсера были отобраны для каждого PAM, отличного от G-PAM, каждый протоспейсер мРНК RFP пронумерован. (B) Нацеливающие на мРНК RFP спейсеры оказывают влияние на нокдаун RFP, тогда как нацеливающие на ДНК спейсеры (кодирующие цепь гена RFP в экспрессирующей плазмиде, обозначенные как спейсеры "rc"), не влияют на экспрессию RFP, число биологических повторений n=3. (C) Количественная оценка нокдауна RFP в E.coli. Три спейсера, нацеливающих на соответственно фланкирующие C-, U- или А-PAM протоспейсеры (9 спейсеров с номерами 5-13 согласно обозначениям ряда (A)) были встроены в мРНК RFP, экспрессия RFP была измерена с помощью поточной цитометрии. Каждая точка на диаграмме рассеяния изображает среднее для трех биологических повторений и соответствует единственному спейсеру. Полосы показывают среднее значение для 4 спейсеров каждого PAM, ошибка показана в виде стандартной ошибки среднего. (D) Анализ кривой роста E.coli. (E) Влияние нацеливания на мРНК RFP на скорость роста трансформированной индуцируемой плазмидой экспрессии RFP и локусом LshC2c2 E.coli с ненацеливающим спейсером, нацеливающим на РНК спейсером (комплементарным некодирующей цепи гена RFP) и нацеливающим на ДНК спейсером (комплементарным кодирующей цепи гена RFP).
[213] Фиг. 124A-124B. Направляемое cr-РНК расщепление оцРНК-мишени активирует неспецифическую РНК-азную активность белка LshC2c2. (A) Схема биохимического анализа неспецифической РНК-азной активности не являющихся мишенью cr-РНК побочных молекул РНК. В дополнение к немаркированному оцРНК-субстрату, являющемуся мишенью РНК CRISP,R в ту же реакцию с целью отследить неспецифическую РНК-азную активность была добавлена вторая оцРНК, меченная на 3'-конце флуоресцентной меткой. (B) В денатурирующем геле показана неспецифическая РНК-азная активность против оцРНК-субстратов, не являющихся мишенью, в присутствии РНК-мишени. Не являющиеся мишенью оцРНК-субстраты не подвергаются расщеплению в отсутствие оцРНК-субстрата, являющегося мишенью cr-РНК.
[214] Фиг. 125. C2c2 является РНК-системой адаптивного иммунитета, предположительно участвующей в абортивной инфекции через программируемую клеточную смерть или индукцию состояния покоя клеток.
[215] Фиг. 126. Секвенирование РНК локуса Leptotrichia shahii, гетерологически экспрессированного в E.coli, и анализ спейсера. Адаптировано по S. Shmakov et al., Discovery and Functional Characterization of Diverse Class 2 CRISPR-Cas Systems, Mol Cell 60, 385-397 (2015). Гетерологическая экспрессия локуса LshC2c2 выявляет процессинг данной последовательности. Вставка: In silico анализ кофолдинга зрелого прямого повтора.
[216] Фиг. 127A-127D. Повторный скрининг с использованием фага MS2 показал воспроизводимое отсутствие 5'-концевого PAM. (A) Сравнение повторностей контроля (без фага) показало воспроизводимость результатов и отсутствие обогащения или обеднения последовательности. (B) Сравнение повторений эксперимента с использованием фага показало как существенное обеднение, так и обогащение популяции, общей для обоих повторений. (C) Логотип последовательностей из обогащенных спейсеров для 5'-конца показывает отсутствие PAM. (D) Частота встречаемости оснований последовательности 5'-конца из обогащенных спейсеров показывает отсутствие PAM.
[217] Фиг. 128A-128G. Скрининг представленности спейсеров в различных PAM с помощью библиотек фага MS2. (A) График типа "ящик с усами", иллюстрирующий распределение частот встречаемости спейсеров, сгруппированных по их 3'-концевой последовательности при условии обработки фагом. Границами ящика являются первый и третий квартили, концы усов соответствуют 1 и 99 перцентилям. ****, p < 0,0001. (B) Множественное сравнение (ANOVA с критерием Тьюки) для всех возможных пар PAM для распределения обработанных фагом спейсеров. Изображены доверительные интервалы для разности средних между сравниваемыми парами PAM. (C) График типа "ящик с усами", иллюстрирующий распределение частот встречаемости спейсеров, сгруппированных по их 3'-концевой последовательности при отсутствии обработки фагом. Границами ящика являются первый и третий квартили, концы усов соответствуют 1 и 99 перцентилям. ****, p < 0,0001. (D) Множественное сравнение (ANOVA с критерием Тьюки) для всех возможных пар PAM для распределения необработанных фагом спейсеров. Изображены доверительные интервалы для разности средних между сравниваемыми парами PAM. (E) Графики кумулятивной частоты встречаемости для log2-нормированного количества спейсеров. Спейсеры разделены соответствующей PAM-последовательностью с целью показать различия в обогащении для каждого распределения PAM. (F) Средняя разность для кумулятивной частоты встречаемости между кумулятивными кривыми при условии обработки и отсутствия обработки фагом. Разности показаны для каждого распределения PAM. (G) Пунктирный контур из (F), показанный крупным планом с целью подчеркнуть различия между обогащениями для различных PAM.
[218] Фиг. 129A-129B. Лучшие результаты скрининга с помощью фага MS2 демонстрируют интерференцию при анализе литических бляшек. (A) Изображения скрининга посредством анализа литических бляшек фага MS2, показывающие сниженное образование литических бляшек для наилучших результатов. Разведения фага были нанесены на бактериальный газон с уменьшением числа бляшкообразующих единиц (БОЕ). Спейсеры-мишени показаны выше изображений; биологические повторности обозначены как BR1, BR2 или BR3. Ненацеливающим контролем является нативный локус белка LshC2c2. (B) Количественная оценка анализа литических бляшек MS2, демонстрирующая интерференцию с участием наилучших результатов. Интерференция была количественно оценена по наибольшему разведению без образования литических бляшек.
[219] Фиг. 130. Анализ литических бляшек фага MS2, подтверждающий наличие 3'-концевого H-PAM. Четыре спейсера для каждого возможного 3'-концевого (A-, G-, C- и U-) PAM были клонированы в вектор pLshC2c2 и проверены на рестрикцию фага MS2 в анализе литических бляшек. Данные изображения показывают значительно сниженное образование литических бляшек для A-, C- и U-PAM и сниженный уровень рестрикции для G-PAM. Фаговые разведения были нанесены бактериальный газон с уменьшением числа бляшкообразующих единиц (БОЕ). Спейсеры-мишени показаны выше изображений; три биологические повторности приведены друг под другом ниже каждой последовательности протоспейсера. Ненацеливающим контролем являются нативный локус белка LshC2c2 и остов pACYC 184.
[220] Фиг. 131A-131D. Очистка белка LshC2c2. (A) Окрашенный кумасси синим акриламидный гель для поэтапной очистки белка LshC2c2. Выраженная полоса непосредственно выше 150 кДа согласуется с размером белка LshC2c2 (171 кДа). (B) Гель-фильтрация для исключения по размеру белка LshC2c2. LshC2c2 элюирован при заданном размере примерно >160 кДа (62,9 мл). (C) Белковые стандарты, использованы для калибровки колонки Superdex 200. BDex=голубой декстран (объем пустот), Ald=альдолаза (158 кДа), Ov=овальбумин (44 кДа), RibA=Рибонуклеаза A (13,7 кДа), Apr=апротинин (6,5 кДа). (D) Кривая калибровки колонки Superdex 200. Kav рассчитана как (элюированный объем - объем пустот)/(геометрический объем колонки - объем пустот). Стандарты изображены на графике в виде логарифмических кривых.
[221] Фиг. 132A-132B. Дальнейшее описание in vitro кинетики расщепления РНК белком LshC2c2. (A) Временной ряд расщепления оцРНК белком LshC2c2, полученный с помощью меченной с 5 '-и 3'-концов мишени 1. (B) Расщепление меченной с 5'-и 3'-концов РНК-мишени 1 с помощью комплекса LshC2c2-CRISPR-РНК на различных этапах серии полулогарифмических разведений.
[222] Фиг. 133. Зависимость расщепления РНК белком LshC2c2 от присутствия ионов металлов. Различные двухвалентные катионы металлов были добавлены в реакционную среду расщепления посредством LshC2c2 с использованием меченой с 5'-конца мишени 1. Существенное усиление расщепления наблюдается только для Mg+2. Для Ca+2 и Mn+2 наблюдается слабое расщепление.
[223] Фиг. 134A-134D. Белок LshC2c2 не показал заметной активности по расщеплению при использовании в качестве субстратов дцРНК, дцДНК или оцДНК. (A) Схема частично двухцепочечной РНК-мишени. Меченную с 5'-конца мишень 1, подвергали отжигу с двумя короткими РНК, комплементарными областям, фланкирующими протоспейсер. Такие частично двухцепочечные РНК служат для более точной проверки нарезания дцДНК, поскольку должны позволять связывание комплекса LshC2c2 с оцРНК. (B) Активность LshC2c2 по расщеплению мишени-дцРНК, показанной в (A) в сравнении с оцРНК-мишенью 1. При использовании в качестве субстрата дцРНК расщепление не обнаружено. (C) Расщепление белком LshC2c2 плазмидной библиотеки дцДНК. Плазмидная библиотека была создана с целью получения семи случайных нуклеотидов в 5'-концевом протоспейсере 14 с целью учета всех возможных условий для расщепления дцДНК. Никакое расщепление не наблюдается для этой библиотеки дцДНК. (D) Проверка варианта оцДНК-мишени 1 на предмет расщепления белков LshC2c2. Расщепление не обнаружено.
[224] Фиг. 135A-135B. LshC2c2 не демонстриурет заметной активности по расщеплению дцДНК-мишеней согласно анализу котрансляционного расщепления. (A) Схема анализа котрансляционного расщепления. C2c2 был инкубирован с элонгационными комплексами РНК-полимеразы E.coli RNA (RNAP) и rNTP, как это описано выше (P. Samai et al., Co-transcriptional DNA and RNA Cleavage during Type III CRISPR- Cas Immunity. Cell 161, 1164-1174 (2015)). (B) Расщепление белком LshC2c2 мишени-ДНК после анализа котрансляционного расщепления. Расщепление не обнаружено.
[225] Фиг. 136. Анализ рестрикции MS2 показывает, что мутанты с единичными мутациями в домене HEPN дезактивируют LshC2c2. Все четыре возможных HEPN с единичными мутациями (R597A, H602A, R1278A и H1283A) были получены в векторе pLshC2c2 с протоспейсером 1. Изображения анализа негативных колоний показывают, что такие мутантные локусы HEPN приводят к образованию негативных колоний, сходному с таковым для ненацеливающего локуса, которое значительно больше, чем для локуса WtC2c2. Разведения фага были высеяны на бактериальный газон с уменьшением числа бляшкообразующих единиц (БОЕ). Спейсеры-мишени показаны выше изображений; биологические повторения обозначены как BR1, BR2 или BR3. Ненацеливающим контролем является нативный локус LshC2c2.
[226] Фиг. 137A-137F. Количественная оценка связывания LshC2c2. (A) Расчет аффинности связывания для комплекса LshC2c2-CRISPR-РНК и оцРНК-мишени. Доля связанного белка была количественно определена с помощью денситометрии на фиг. 413, KD была вычислена по изотерме связывания. (B) Расчет аффинности связывания комплекса LshC2c2 с CRISPR-РНК для мутанта по HEPN R1278A в сравнении с мишенью-оцРНК. Доля связанного белка была количественно определена с помощью денситометрии на фиг. 4E, KD была вычислена по изотерме связывания. (C) Анализ способом сдвига электрофоретической подвижности для мутанта по HEPN белка LshC2c2 R1278A в сравнении с мишенью-оцРНК в отсутствие cr-РНК. В реакционную среду был добавлен ЭДТА. (D) Анализ способом сдвига электрофоретической подвижности для cr-РНК в сравнении с оцРНК-мишенью. В реакционную среду был добавлен ЭДТА. (E) Расчет сродства связывания LshC2c2 мутанта по HEPN R1278A с оцРНК-мишенью в сравнении с мишенью-оцРНК. Доля связанного белка была количественно определена с помощью денситометрии на фиг. S12C, KD была вычислена по изотерме связывания. (F) Расчет сродства связывания CRISPR-РНК с оцРНК-мишенью. Доля связанной CRISPR-РНК была количественно определена с помощью денситометрии на фиг. S12D, KD была вычислена по изотерме связывания.
[227] Фиг. 138. Анализ рестрикции в MS2 с целью выявить влияние единичных и двойных нарушений комплементарности на активность LshC2c2. Вектор pLshC2c2 с протоспейсером 41 был модифицирован с целью получения ряда единичных нарушений комплементарности, как это показано на рисунке. Изображения анализа негативных колоний, выполненного с целью проверки таких спейсеров с нарушением комплементарности и выявившего уменьшение образования для спейсеров с единичными нарушениями комплементарности наравне с полностью комплементарным спейсером. Спейсеры с двойными нарушениями комплементарности демонстрируют усиленное образование негативных колоний в случае если это нарушение локализовано в последовательности затравки в середине последовательности спейсера. Разведения фага высевали на чашках с бактериями с последовательным уменьшением бляшкообразующих единиц (БОЕ). Мишени спейсера приведены выше изображений; биологические повторения обозначены как BR1, BR2 или BR3. Ненацеливающим контролем является нативный локус LshC2c2.
[228] Фиг. 139. Мутант по HEPN белка LshC2c2 был проверен на активность нацеливания на мРНК RFP. Вектор pLshC2c2 с протоспейсером 36 был модифицирован таким образом, чтобы были получены единичные мутации по HEPN R597A и R1278A (по одной в каждом из доменов HEPN). Эти мутации привели к небольшому, но обнаружимому нокдауну RFP, согласно данным поточной цитометрии для E.coli.
[229] Фиг. 140A-140B. Биохимическое описание сопутствующего эффекта расщепления. (A) LshCc2 был инкубирован с cr-РНК, нацеливающей на протоспейсер 14, в присутствии и в отсутствие немеченной мишени-оцРНК 1 (содержит протоспейсер 14). В присутствии мишени 1 белка LshC2c2 наблюдается существенное расщепление флуоресцентно меченых некомплементарных мишеней 6-9. (B) Побочная активность мутанта по HEPN в сравнении с WT C2c2. Белки были инкубированы с комплементарной к протоспейсеру 14 cr-РНК в присутствии и в отсутствии немеченых гомополимеров-мишеней 2 или 3 (обе содержат протоспейсер 14). Сопутствующий эффект белков с мутациями по HEPN в отношении флуоресцентно меченой некомплементарной мишени 8 более не наблюдается.
[230] Фиг. 141A-141B. Подавление роста коррелирует с нокдауном RFP в экспрессирующих C2c2 и cr-РНК, нацеленную на RFP, клетках. На фиг. 141A представлено влияние на рост экспрессии RFP вызываемой в E.coli, экспрессирующей C2c2 и cr-РНК, нацеленную на RFP. RFP был экспрессирован с использованием индуцируемой ангидротетрациклином (aTc) системы экспрессии гена. Клеточные культуры были обработаны aTc в указанных концентрациях. Увеличение экспрессии aTc подавляло роста клеток. На фиг. 141B представлено влияние факта замены ненацеленной cr-РНК на нацеленную на RFP cr-РНК на рост индуцибельной экспрессии RFP.
[231] Фиг. 142A-142D. C2c2 предпочтительно расщепляет поли(U)-участки. Расщепление оцРНК белком C2c2 было исследовано с использованием субстрата-оцРНК, содержащего либо не содержащего поли(U)-участки. Меченая на конце оцРНК была инкубирована с C2c2 и cr-РНК с установлением продуктов расщепления. Субстрат-РНК, содержащий поли(U)-участки, расщеплялся эффективно. Расщепление субстрата носило зависимый от cr-РНК характер, как следует из отсутствия продуктов расщепления в присутствии C2c2, но не cr-РНК. Ряды A и B: субстрат, меченый с 3'-конца. Ряды C и D: субстрат, меченый с 5'-конца. Ряды В и D указывают на эффективность и специфичность расщепления субстрата при увеличении концентрации субстрата.
[232] Фиг. 143A-143B. Расщепление РНК-мишени, имеющей нарушение комплементарности cr-РНК в зависимости от ее положения. ОцРНК-мишень была подвергнута воздействию cr-РНК, содержащей двойные (ряд A) или тройные (ряд B) нарушения комплементарности, а также LshC2c2. Продукты реакции расщепления были разделены припомощи электрофореза.
[233] Фиг. 144. Расщепление РНК-мишени чувствительно к мутациям и делециям в области 3'-концевых прямых повторов. ОцРНК-мишень была подвергнута воздействию LshC2c2 и cr-РНК, содержащей нарушения комплементарности или делеции, введенные с целью нарушить вторичную структуру в области прямых повторов (DR). Продукты реакций расщепления были разделены с помощью электрофореза.
[234] Фиг. 145A-145B. C2c2 и MS2-CRISPR-РНК могут связывать оцРНК. Фиг. 145A: определение связывания LshC2c2(R1278A), являющегося дефектным в отношении расщепления субстрата. Фиг. 145B: LshC2c2(R1278A) и MS2-CRISPR-РНК были инкубированы с увеличением количества меченой оцРНК 10 (левый ряд) или меченой обратно комплементарной (RC) оцРНК 10 (правый ряд) (последовательность оцРНК 10 см. в таблице В).
[235] Фиг. 146. Комплекс C2C2 не связывает оцДНК. LshC2c2(R1278A) и MS2-CRISPR-РНК были инкубированы с увеличением количества оцДНК 10 (левый ряд) или обратно комплеметарной оцДНК 10 (RC) (правый ряд) (последовательность оцРНК 10 см. в таблице В).
[236] Чертежи приведены в настоящем описании исключительно в иллюстративных целях и не обязательно выполнены с соблюдением масштаба.
ДЕТАЛЬНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
[237] В целом, понятие "система CRISPR-Cas или CRISPR", используемое также в предшествующих документах, таких как W0 2014/093622 (PCT/US2013/074667), используется для совместного обозначения транскриптов и других компонентов, участвующих в экспрессии или управлении активностью CRISPR-ассоциированных ("Cas") генов, включая последовательности, кодирующие ген Cas, последовательность tracr (трансактивирующая CRISPR) (например, tracr-РНК или активная частичная tracr-РНК), последовательность tracr-помощника (включащая "прямой повтор" и процессируемый tracr-РНК частичный прямой повтор в составе эндогенной системы CRISPR), направляющую последовательность (также называемую "спейсером" в составе эндогенной системы CRISPR) или "молекулу(ы) РНК", как этот термин использован в настоящем описании (например, молекулу(ы) РНК, направляющую Cas, такие как Cas9, например, CRISPR-РНК и трансактивирующую (tracr) РНК или одиночную направляющую РНК (sgRNA) (химерная РНК)) или другие последовательности и транскрипты локуса CRISPR. В целом, система CRISPR характеризуется компонентами, которые способствуют формированию комплекса CRISPR на участке последовательности-мишени (в составе эндогенной системы CRISPR также называемый протоспейсером). Если белком CRISPR является белок C2c2, tracr-РНК не требуется.
[238] В контексте формирования комплекса CRISPR, "последовательность-мишень" означает последовательность, для которой целенаправленно разработана комплементарная направляющая последовательность, причем гибридизация между последовательностью-мишенью и направляющей последовательностью способствует образованию комплекса CRISPR. Последовательность-мишень может включать любой полинуклеотид, в том числе полинуклеотиды РНК или ДНК. В некоторых вариантах осуществления изобретения последовательность-мишень расположена в ядре или цитоплазме клетки. В некоторых вариантах осуществления изобретения прямые повторения могут быть идентифицированы in silico путем поиска повторяющихся мотивов, которые удовлетворяют любому или всем из следующих критериев: 1. обнаружены в пределах области геномной последовательности размером 2 т.п.н., фланкирующей локусы CRISPR II типа; 2. длина от 20 до 50 п.н.; и 3. разделяющие их промежутки длиной 20-50 п.н. В некоторых вариантах осуществления изобретения могут быть использованы любые 2 из этих критериев, например, 1 и 2, 2 и 3 или 1 и 3. В некоторых вариантах осуществления изобретения могут быть использованы все 3 критерия.
[239] В вариантах осуществления изобретения такие понятия как направляющая последовательность и направляющая РНК, т.е. РНК, способная направлять Cas к целевому локуса генома, используются взаимозаменяемо в предшествующих цитируемых документах, таких как W0 2014/093622 (PCT/US 2013/074667). В целом, направляющей последовательностью является любая полинуклеотидная последовательность, имеющая комплементарность к полинуклеотиду-мишени, достаточную для гибридизации с последовательностью-мишенью и направления последовательности на прямое специфическое связывание комплекса CRISPR с последовательностью-мишенью. В некоторых вариантах осуществления изобретения уровень комплементарности между направляющей последовательностью и соответствующей ей последовательностью-мишенью при условии оптимального выравнивания с использованием надлежащего алгоритма выравнивания близок или превосходит значения 50%, 60%, 75%, 80%, 85%, 90%, 95%, 97,5%, 99% или выше. Оптимальное выравнивание может быть определено с использованием любого надлежащего алгоритма для выравнивания последовательностей включая, но не ограничиваясь следующими: алгоритм Смита-Ватермана, алгоритм Нидлмана-Вунша, алгоритмы, основанные на преобразовании Барроуза-Уилера (например, Burrows Wheeler Aligner), ClustalW, Clustal X, BLAST, Novoalign (Novocraft Technologies; доступен на www.novocraft.com), ELAND (Illumina, Сан-Диего, Калифорния, США), SOAP (доступен на soap.genomics.org.cn) и Maq (доступен на maq.sourceforge.net). В некоторых вариантах осуществления направляющая последовательность имеет длину, примерно равную или превышающую 5, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 75 или более нуклеотидов. В некоторых вариантах осуществления направляющая последовательность имеет длину, меньшую 75, 50, 45, 40, 35, 30, 25, 20, 15, 12 или менее нуклеотидов. Предпочтительная длина направляющей последовательности составляет 10-30 нуклеотидов. Способность направляющей последовательности направлять специфически определяемое последовательностью связывание комплекса CRISPR с последовательностью-мишенью может быть оценена любой подходящей методикой. Например, компоненты системы CRISPR, достаточные для формирования комплекса CRISPR, включая проверяемую направляющую последовательность, могут быть доставлены в клетку-хозяина, содержащую соответствующую последовательность-мишень, в том числе путем трансфекции с использованием векторов, кодирующих компоненты последовательности CRISPR с последующей количественной оценкой локализации предпочтительного расщепления последовательности-мишени, в частности, с использование анализа, как описано в настоящем описании. Сходным образом расщепление последовательности нуклеотида-мишени может быть оценено in vitro при использовании последовательности-мишени, компонентов комплекса CRISPR, включая проверяемую направляющую последовательность и направляющую последовательность-контроль, отличную от тестируемой направляющей последовательности при сравнении уровня или скорости расщепления последовательности-мишени между реакциями с участием тестируемой и контрольной направляющих последовательностей. Другие способы анализа также допустимы и могут быть выбраны специалистом в данной области.
[240] В классических системах CRISPR-Cas уровень комплементарности между последовательностью направляющей молекулы и соответствующей ей последовательностью-мишенью может быть примерно равен или превышать 50%, 60%, 75%, 80%, 85%, 90%, 95%, 97,5%, 99% или 100%; направляющая молекула или РНК или sg-РНК могут быть иметь длину, примерно равную или превышающую 5, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 75 или более нуклеотидов; либо направляющая молекула или РНК или sg-РНК могут иметь длину, меньшую чем приблизительно 75, 50, 45, 40, 35, 30, 25, 20, 15, 12 или менее нуклеотидов. Однако в одном из своих аспектов изобретение призвано уменьшить нецелевые взаимодействия, например, уменьшить взаимодействие направляющей молекулы с молекулой-мишенью с низкой комплементраностью. Действительно, в примерах показано, что в изобретение вовлекает мутации, делающие систему CRISPR-Cas способной различать последовательности-мишени и последовательности, не являющиеся мишенями, с комплементарностью более 80% до примерно 95%, например, с комплементарностью 83%-84%, 88-89% или 94-95% (к примеру, различая молекулу-мишень, имеющую 18 нуклеотидов молекулы, не являющейся мишенью, длиной в 18 нуклеотидов с 1, 2 или 3 нарушениями комплементарности). В соответствии с этим, в контексте настоящего изобретения уровень комплементарности между направляющей последовательностью и соответствующей ей молекулой-мишенью превышает 94,5%, 95%, 95,5%, 96%, 96,5%, 97%, 97,5%, 98%, 98,5%, 99%, 99,5%, 99,9% или 100%. Для молекулы, не являющейся мишенью, он составляет менее 100%, 99,9%, 99,5%, 99%, 99%, 98,5%, 98%, 97,5%, 97%, 96,5%, 96%, 95,5%, 95%, 94,5%, 94%, 93%, 92%, 91%, 90%, 89%, 88%, 87%, 86%, 85%, 84%, 83%, 82%, 81% или 80% комплементарности между ее последовательностью и направляющей молекулой, причем предпочтительно, чтобы не являющаяся мишенью молекула имела комплементарность последовательности 100%, 99,9%, 99,5%, 99%, 99%, 98,5%, 98%, 97,5%, 97%, 96,5%, 96%, 95,5%, 95% или 94,5% с направляющей молекулой.
[241] В некоторых вариантах осуществления изобретения направленные изменения эффективности расщепления могут быть достигнуты за счет введения нарушений комплементарности, например 1 или более нарушений комплементарности, к примеру 1 или 2 нарушения комплементарности между последовательностью спейсера и последовательностью-мишенью, включая положение нарушения комплементарности вдоль спейсера/мишени. Чем более отдаленное от концов (т.е. не 3'- или 5'-концевое) положение занимает, в частности, двойное нарушение комплементарности, тем больше изменится эффективность расщепления. В соответствии с этим, выбирая локализацию нарушения комплементарности на последовательности спейсера, можно направленно изменять эффективность расщепления. К примеру, если желательно расщепление меньше чем 100% молекул-мишеней (например, в популяции клеток), возможно введение 1 или больше, предпочтительно 2-х, нарушений комплементарности между спейсером и последовательностью-мишенью. Чем более центральное положение имеет нарушение комплементарности, тем ниже процент расщепленных молекул.
[242] Согласно изобретению, описанные в настоящем описании способы подразумевают индукцию одной или более модификаций нуклеотидов в эукариотической клетке (in vitro, т.е. в изолированной эукариотической клетке), как описано в настоящем описании, включая доставку в клетку вектора. Такая мутация(и) может подразумевать вставку, удаление или замену одного или более нуклеотидов в каждой последовательности-мишени указанных клеток посредством направляющей(направляющих) РНК или sg-РНК. Мутации могут включать инсерцию, делецию или замену 1-75 нуклеотидов в каждой последовательности-мишени указанной клетки(клеток) с использованием направляющей(их) РНК. Такие мутации могут включать инсерцию, делецию или замену 1, 5, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50 или 75 нуклеотидов в каждой последовательности-мишени указанной клетки(клеток) с использованием направляющей(их) РНК. Такие мутации могут включать инсерцию, делецию или замену 5, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50 или 75 нуклеотидов в каждой последовательности-мишени указанной клетки(клеток) с использованием направляющей(их) РНК. Такие мутации могут включать инсерцию, делецию или замену 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50 или 75 нуклеотидов в каждой последовательности-мишени указанной клетки(клеток) с использованием направляющей(их) РНК. Такие мутации могут включать инсерцию, делецию или замену 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, или 75 нуклеотидов в каждой последовательности-мишени указанной клетки(клеток) с использованием направляющей(их) РНК. Такие мутации могут включать инсерцию, делецию или замену 40, 45, 50, 75, 100, 200, 300, 400 или 500 нуклеотидов в каждой последовательности-мишени указанной клетки(клеток) с использованием направляющей(их) РНК.
[243] Для минимизации токсичности и побочных эффектов важно управлять концентрациями доставленных мРНК или белка Cas и направляющей РНК. Оптимальные концентрации мРНК или белка Cas и направляющей РНК могут быть определены путем проверки различных концентраций в клеточной или животной (эукариотического животного, отличного от человека) и применения глубокого секвенирования для анализа протяженности модификации на потенциальном геномном локусе, не являющемся целевым.
[244] Как правило, в контексте эндогенной системы CRISPR, образование комплекса CRISPR (содержащего направляющую последовательность, гибридизованную с последовательностью-мишенью и образовавшую комплекс с одним или более белками Cas), приводит к расщеплению непосредственно в последовательности-мишени или вблизи нее (например, на расстоянии в 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 50 или более пар оснований).
[245] Предпочтительно, чтобы молекула нуклеиновой кислоты, кодирующая Cas, представляла сбой кодон-оптимизированную последовательность кодонам Cas. Примером оптимизированной по кодонам последовательности в данном случае является последовательность, оптимизированная для экспрессии в эукариоте, например, человеке (т.е. являющаяся оптимизированной для экспрессии в человеке) или другом эукариоте, животном или млекопитающем, как обсуждается в настоящем описании; см, например, кодон-оптимизированную для человека последовательность SaCas9 в WO 2014/093622 (PCT/US2013/074667). Хотя это будет является предпочтительным, также будет понятно, что возможны другие примеры и оптимизация кодонов для клеток-хозяев, отличных от клеток человека, или оптимизация кодонов для конкретных органов известна. В некоторых вариантах осуществления, кодирующая фермент последовательность кодирует Cas и кодон-оптимизирована для экспрессии в конкретных клетках, например, эукариотических клетках. Такие эукариотические клетки могут принадлежать или быть выделены из конкретного организма, в частности млекопитающего, включая, но не ограничиваясь этим: человека, отличного от человека эукариота, животного или млекопитающего, как описано в настоящем описании, например, мыши, крысы, кролика, собаки, домашнего скота или отличного от человека млекопитающего или примата. В некоторых вариантах осуществления, способы модификации генетической принадлежности зародышевой линии человека и/или способы модификации генетической принадлежности животных, которые с большой долей вероятности могут причинить им страдания без получения существенной медицинской пользы для человека или животных, а также получаемые в результате таких способов животные, могут быть исключены. В целом под оптимизацией кодонов понимается процесс изменения последовательности с целью усиления экспрессии в клетках заданного организма путем замены по меньшей мере одного кодона (например, примерно или более 1, 2, 3, 4, 5, 10, 15, 20, 25, 50 или большего количества кодонов) нативной последовательности на кодоны, которые более часто или наиболее часто используются в генах таковой клеток целевого организма при сохранении исходной аминокислотной последовательности. Различные виды демонстрируют конкретное смещение в частоте встречаемости отдельных кодонов для отдельных аминокислот. Смещение частоты кодонов (различие в использовании кодонов между различными организмами) зачастую коррелирует с эффективностью трансляции матричной РНК (мРНК), которая в свою очередь считается зависящей, в числе прочего, от свойств транслируемых кодонов и доступности отдельных молекул транспортной РНК (тРНК). Преобладание конкретных тРНК в клетке обычно соответствует тем кодонам, которые используются наиболее часто при синтезе пептидов. В соответствии с этим, гены могут быть разработаны с учетом оптимизации кодонов, делающей экспрессию генов оптимальной в некотором организме. Таблицы используемых кодонов доступны, к примеру, в "Базе данных использования кодонов" ("Codon Usage Database"), доступной по ссылке www.kazusa.orjp/codon/, причем такие таблицы могут быть адаптированы рядом способов. См. Nakamura, Y., et al. "Codon usage tabulated from the international DNA sequence databases: status for the year 2000" Nucl. Acids Res. 28:292 (2000). Также доступны компьютерные алгоритмы оптимизации кодонов для конкретной последовательности и экспрессии в конкретной клетке-хозяине, такие как Gene Forge (Aptagen; Джакобус, Пенсильвания, США). В некоторых вариантах осуществления один или более кодонов (например, 1, 2, 3, 4, 5, 10, 15, 20, 25, 50 или более, либо все кодоны) в последовательности, кодирующей Cas, соответствуют наиболее часто используемым кодонам для определенной аминокислоты.
[246] В некоторых вариантах осуществления изобретения способы, как описано в настоящем описании, могут включать доставку Cas в трансгенную клетку, в которой одна или более нуклеиновых кислот, кодирующих одну или более молекул направляющих РНК, доставленных или непосредственно введенных, связаны в клетке с регуляторным элементом, содержащим промотор одного или более генов-мишеней. Термин "трансгенная по Cas клетка", как используют в настоящем описании, относится к клетке, в частности, эукариотической клетке, в геном которой встроен ген Cas. Природа, тип или происхождение такой клетки не ограничивается особым образом, согласно данному изобретению. Также способ введения трансгена Cas в клетку может различаться и являться любым из способов, доступных в данной области. В некоторых вариантах осуществления изобретения трансгенная клетка Cas получена путем введения трансгена Cas в выделенную клетку. В некоторых других вариантах осуществления изобретения трансгенная клетка Cas может быть получена путем выделения клеток из трансгенного организма Cas. В качестве не ограничивающего примера, трансгенная клетка Cas, как используют в настоящем описании, может быть получена из трансгенного по Cas эукариотического организма, такого как эукориотический организм с встроенным Cas. См. WO 2014/093622 (PCT/US 13/74667), включенную в настоящее описание в качестве ссылки. Способы публикации патентов США № 20120017290 и 20110265198, закрепленных за Sangamo BioSciences, Inc., направленные на нацеливание на локус Rosa, могут быть изменены для использования системы CRISPR Cas данного изобретения. Способы публикации патента США № 20130236946, закрепленных за Cellectis, направленные на нацеливание на локус Rosa, могут также быть изменены для использования системы CRISPR-Cas по настоящему изобретению. В качестве следующего примера приводится ссылка на статью Platt et. al. (Cell; 159(2):440-455 (2014)) с описанием мыши со вставкой Cas9 мыши, включенную в настоящее описание в качестве ссылки. Трансген Cas, кроме того, может включать кассету Lox-Stop-polyA-Lox(LSL), что делает экспрессию Cas управляемой с помощью рекомбиназы Cre. Альтернативно, клетка с трансгенным Cas может быть получена путем введения трансгена Cas в выделенную клетку. Системы доставки трансгенов хорошо известны в данной области. Примером может служить доставка трансгена Cas, напримр, в эукариотическую клетку посредством вектора (например, AAV, аденовируса, лентивируса) и/или частицы и/или наночастицы, как также описано в другой части настоящего описания.
[247] Для квалифицированного специалиста будет понятно, что клетка, такая как трансгенная по Cas клетка в используемом в настоящем описании значении, может содержать дальнейшие изменения генома помимо наличия встроенного гена Cas или мутаций, являющихся результатом последовательность-специфического действия Cas, в комплексе с РНК, способной направлять Cas к локусу-мишени, например, такие как одна или более онкогенных мутаций, как в качестве неограничивающего примера описано в Platt et al. (2014), Chen et al., (2014), или Kumar et al. (2009).
[248] В некоторых вариантах осуществления последовательность Cas объединена с одной или более последовательностями сигнала ядерной локализации (NLS), в частности примерно с или более чем примерно с 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS. В некоторых вариантах осуществления Cas содержит примерно или более 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS на N-конце или вблизи него, примерно или более 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS на C-конце или их комбинацию (например, ноль или по меньшей мере один или более NLS на N-конце и ноль или по меньшей мере один или более NLS на С-конце). В случае присутствия более одного NLS каждый из них может быть выбран независимо от других, в частности, отдельный NLS может быть представлен более чем одной копией и/или в комбинации с другим одним или более NLS, представленного одной или более копиями. В предпочитаемом варианте осуществления Cas содержит не более 6 NLS. В некоторых вариантах осуществления NLS считается N- или C-концевым, если ближайшая к концу аминокислота NLS находится на расстоянии 1, 2, 3, 4, 5, 10, 15, 20, 25, 30, 40, 50 или более аминокислот полипептидной цепи от N- или C-конца. Неограничивающие примеры NLS включают последовательности NLS, полученные на основе: NLS большого T-антигена вируса SV40, имеющего аминокислотную последовательность PKKKRKV (SEQ ID NO: X); NLS из нуклеоплазмина (например, состоящая из двух частей NLS нуклеоплазмана с последовательностью KRPAATKKAGQAKKKK) (SEQ ID NO: X); NLS c-myc, имеющего аминокислотную последовательность PAAKRVKLD (SEQ ID NO: X) или RQRRNELKRSP(SEQ ID NO: X); NLS hRNPA1 M9, имеющего последовательность NQ S SNFGPMKGGNFGGRS SGPYGGGGQYFAKPRNQGGY (SEQ ID NO: X); последовательности RMRIZFKNKGKDTAELRRRRVEVSVELRKAKKDEQILKRRNV (SEQ ID NO: X) из домена импортина-альфа; последовательности VSRKRPRP (SEQ ID NO: X) и PPKKARED (SEQ ID NO: X) T-белка миомы; последовательности POPKKKPL (SEQ ID NO: X) p53 человека; последовательности SALIKKKKKMAP (SEQ ID NO: X) c-abi IV мыши, последовательности DRLRR (SEQ ID NO: X) и PKQKKRK (SEQ ID NO: X) вируса гриппа NS1; последовательности RKLKKKIKKL (SEQ ID NO: X) антигена дельта вируса гепатита; последовательности REKKKFLKRR (SEQ ID NO: X) белка Mx1 мыши; последовательности KRKGDEVDGVDEVAKKKSKK (SEQ ID NO: X) поли(АДФ-рибоза) полимеразы человека и последовательности RKCLQAGMNLEARKTKK (SEQ ID NO: X) рецепторов глюкокортикоидных стероидных гормонов (человека). В целом, такая одна или более NLS имеют достаточную силу для того, чтобы вызвать накопление Cas в заметных количествах в ядре эукариотической клетки. В целом сила активности ядерной локализации может определяться рядом NLS в белках Cas, конкретным(и) использованным(и) NLS или комбинацией этих факторов. Например, поддающаяся обнаружению метка может быть связана с Cas таким образом, чтобы визуализировать локализацию в клетке, в частности, за счет сочетания способов определения локализации ядра (например, специфичный ядерный краситель, такой как DAPI). Клеточные ядра могут быть также выделены из клеток, содержимое которых может затем быть проанализировано с помощью любого подходящего способы обнаружения белков, такого как иммуногистохимия, вестерн-блоттинг или анализ активности ферментов. Накопление в ядре может быть определено косвенно, в частности, с помощью анализа эффекта образования комплексов CRISPR (например, анализ расщепления ДНК или мутаций в последовательности-мишени, или анализ изменения экспрессии генов вследствие образования комплексов CRISPR и/или активности ферментов Cas), в сравнении с контролем, не подвергнутым воздействию Cas или комплекса или подвергнутым действия Cas, лишенного одной или более NLS, или сигнала ядерного экспорта (NES). В некоторых вариантах осуществления изобретения с белком Cas могут быть связаны другие метки локализации, неограничивающим примером локализации Cas в конкретных областях клетки могут быть органеллы, такие как митохондрии, пластиды, хлоропласты, везикулы, комплекс Гольджи, (ядерные или клеточные) мембраны, рибосомы, ядрышко, эндоплазматический ретикулум (ER), цитоскелет, вакуоли, центросомы, нуклеосомы, гранулы, центриоли и т.д.
[249] В некоторых аспектах изобретение включает такие векторы, например, для доставки или введения в клетку Cas и/или РНК, способной направлять Cas к локусу-мишени (т.е. направляющей РНК), а также для воспроизведения этих компонентов (например, в прокариотических клетках). В используемом в настоящем описании значении "вектор" представляет собой инструмент, позволяющий или облегчающий перенос некоторого объекта из одного окружения в другое. Он представляет собой репликон, такой как плазмида, фаг или космида, в который может быть вставлен другой участок ДНК таким образом, чтобы добиться репликации вставленного участка. В целом вектор способен к репликации при условии ассоциации с надлежащими элементами контроля. В целом понятие "вектор" обозначает молекулу нуклеиновой кислоты, способную транспортировать другую нуклеиновую кислоту, с которой она связана. Неограничивающими примерами векторов являются молекулы нуклеиновых кислот, являющихся одноцепочечными, двухцепочечными или частично двухцепочечными; молекулы нуклеиновых кислот, имеющие один или более свободных концов, не имеющие свободных концов (например, кольцевые); молекулы нуклеиновых кислот, включающие ДНК, РНК или одновременно и ДНК и РНК и другие известные в данной области разновидности полинуклеотиды. Одним из типов векторов является "плазмида", представляющая собой кольцевую двухцепочечную петлю ДНК, в которую могут быть вставлены дополнительные участки ДНК, в том числе с использованием стандартных способов клонирования. Другим типом вектора является вирусный вектор, содержащий полученную из вирусной ДНК или РНК последовательность, необходимую для упаковки в вирус (например, ретровирусы, дефективные в отношении репликации ретровирусы, аденовирусы, аденовирусы, ретровирусы и аденоасоциированные вирусы (AAV, AAV) с нарушенной репликацией). Вирусные векторы также включают полинуклеотиду, помещенную в вирус с целью трансфекции в клетку-хозяина. Некоторые векторы способны к автономной репликации в клетке хозяина, в которую они введены (например, бактериальные векторы, имеющие бактериальный ориджин репликации и эписомные векторы млекопитающих). Другие векторы (например, векторы млекопитающих, отличные от эписомных) интегрируются в геном клетки хозяина при введении в клетку хозяина и вследствие этого реплицируются вместе с геномом хозяина. Более того, некоторые векторы способны управлять экспрессией генов, с которыми они непосредственно связаны. Такие векторы называются в настоящем описании "экспрессирующими векторами". Распространенной формой экспрессии вспомогательных векторов в способах рекомбинантных ДНК часто являются плазмиды.
[250] Рекомбинантные экспрессирующие векторы могут включать нуклеиновую кислоту по изобретению в форме, подходящей для экспрессии нуклеиновой кислоты в клетке-хозяине, что означает, что рекомбинантные экспрессирующие векторы включают один или более регуляторных элементов, которые могут быть подобраны на основе клеток-хозяев, которые будут использоваться для экспрессии, т.е. функционально связаны с последовательностью нуклеиновых кислот, которая будет экспрессирована. В рекомбинантном векторе экспрессии "непосредственно связанный" должно означать, что представляющая интерес нуклеотидная последовательность связана с регуляторным элементом(элементами) таким образом, что обеспечивает экспрессию нуклеотидной последовательности (например, в системе транскрипции/трансляции in vitro или в клетке-хозяине, когда вектор введен в клетку-хозяина). Относительно рекомбинации и способов клонирования, упоминается заявка на патент США № 10/815730, опубликованная 2 сентября 2004 года как US 2004-0171156 A1, содержание которой включено в настоящее описание в качестве ссылки в полном объеме.
[251] Вектор(ы) может включать регуляторный элемент(ы), например, промотор(ы). Вектор(ы) может включать последовательности, кодирующие белки Cas и/или единственную направляющую последовательность РНК, однако возможно также по меньшей мере 3, или 8, или 16, или 32, или 48, или 50 последовательностей, кодирующих направляющие последовательности РНК (например, sg-РНК), например 1-2, 1-3, 1-4 1-5, 3-6, 3-7, 3-8, 3-9, 3-10, 3-8, 3-16, 3-30, 3-32, 3-48, 3-50 РНК (например, sg-РНК). Единственный вектор может содержать промотор для каждой РНК (например, sg-РНК), предпочтительно, когда он содержит приблизительно 16 молекул РНК; когда единственный вектор предусматривает больше, чем 16 молекул РНК, один или более промотор(ов) могут управлять экспрессией больше чем одной молекулы РНК, например, когда вектор содержит 32 молекулы РНК, каждый промотор управляет экспрессией двух молекул РНК, и когда вектор содержит 48 молекул РНК, каждый промотор управляет экспрессией трех молекул РНК. С помощью простой арифметики и устоявшихся протоколов клонирования и руководства в данной заявке квалифицированный специалист в данной области может применять изобретение на практике в отношении РНК для подходящего иллюстративного вектора, такого как AAV, и подходящего промотора, такого как промотор U6. Например, предел упаковки AAV составляет ~4,7 т.п.н. Длина единичной конструкции U6-гРНК (включая участки рестрикции для клонирования) составляет 361 п.н. Следовательно, квалифицированный специалист может без труда совместить приблизительно 12-16, например, 13 кассет U6-гРНК в одном векторе. Данная конструкция может быть собрана любыми подходящими способами, такими как стратегия "золотых ворот", используемая для сборки TALE (http://www.genome-engineering.org/taleffectors/). Квалифицированный специалист может также использовать стратегию "тандемного направления" для увеличения числа структур U6-гРНК приблизительно в 1,5 раза, например, от 12-16, например, 13, до приблизительно 18-24, например, приблизительно 19 гРНК U6. Следовательно, квалифицированный специалист в данной области может без труда получить приблизительно 18-24, например, приблизительно 19 структур промотор-РНК, например, U6-гРНК, в единственном векторе, например, векторе AAV. Следующим способом увеличения числа промоторов и РНК в векторе является использование единственного промотора (например, U6) для экспрессии последовательности РНК, разделенных последовательностями, способными к расщеплению. Следующим шагом для увеличения числа структур промотор-РНК в векторе является экспрессия последовательности структур промотор-РНК, разделенных способными к расщеплению последовательностями, в интроне кодирующей последовательности или гене. В этом случае предпочтительно использовать промотор полимеразы II, который может иметь увеличенную экспрессию и обеспечивать транскрипцию длинной РНК, тканеспецифическим образом (см., например, http://nar. oxfordjournals.org/content/34/7/e53.short, http://www.nature.com/mt/journal/v16/n9/abs/mt2008144a.html). В предпочтительном варианте осуществления изобретения в вектор AAV может быть упакован тандем U6-gРНК, нацеленный приблизительно на вплоть до 50 генов. Соответственно, исходя из знаний в данной области и руководства в настоящей заявке, квалифицированный специалист может создавать и использовать вектор(ы), например, единичный вектор, экспрессирующий множественные РНК или другие направляющие молекулы под контролем, оперативно или функционально связанный с одним или более промоторами, особенно для числа РНК или других направляющих молекул, обсуждаемого в настоящем описании, без какого-либо излишнего экспериментирования.
[252] Последовательности, кодирующие направляющие РНК и/или последовательности, кодирующие белки Cas, могут быть функционально или непосредственно связаны с регуляторным элементом(элементами) и, следовательно, регуляторный элемент(элементы) управляет их экспрессией. Промотор(промоторы) может быть конститутивным промотором (промоторами) и/или временным промотором(промоторами), и/или индуцируемым промотором(промоторами), и/или тканеспецифическим промотором(промоторами). Промотор может быть выбран из группы, состоящей из РНК-полимераз: pol I, pol II, pol III, T7, U6, H1, ретровирусного LTR-промотора (LTR) вируса саркомы Рауса (RSV), промотора цитомегаловируса (CMV), промотора вируса SV40, промотора дигидрофолатредуктазы, промотора β-актина, промотора фосфоглицераткиназы (PGK) и промотора EF1α. Предпочтительным промотором является промотор U6.
[253] Локусы системы CRISPR-Cas насчитывают более 50 семейств генов и не имеют строго универсальных генов. Следовательно, для локусов системы CRISPR-Cas ни одно эволюционное дерево не будет единственно верным, и для идентификации новых семейств генов необходим многоплановый подход. На данный момент для 395 профилей 93 белков Cas исчерпывающе идентифицированы гены cas. Для классификации систем CRISPR-Cas используют профили генов и характерные особенности архитектуры локусов. Новая классификация систем CRISPR-Cas предложена на фиг. 1A И 1B. Класс 1 включает многосубъединичные эффекторные комплексы cr-РНК (Cascade), и Класс 2 включает односубъединичные эффекторные комплексы cr-РНК (Cas9-подобные). На фиг. 2 изображена молекулярная организация систем CRISPR-Cas. На фиг. 3 изображена структура эффекторных комплексов I и III типов: общая архитектура/общее происхождение прослеживается несмотря на обширную дивергенцию последовательностей. На фиг. 4 CRISPR-Cas изображена как систему, основанную на мотивах распознавания РНК (RRM). На фиг. 5 изображена филогения белка Cas1, показан главный аспект эволюции системы CRISPR-Cas - рекомбинация эффекторных модулей cr-РНК в целях адаптации. На фиг. 6 представлен перечень систем CRISPR-Cas, а именно распространение типов/подтипов систем CRISPR-Cas среди архей и бактерий.
[254] Действие системы CRISPR-Cas обычно делится на три этапа: (1) адаптация или интеграция спейсера, (2) процессинг первичного транскрипта локуса CRISPR (пре-cr-РНК) и созревание cr-РНК, которая включает спейсер и вариабельные регионы, соответствующие фрагментам CRISPR-повторов на 5'- и 3'-концах, и (3) РНК- или ДНК-интерференция. Два белка, Cas1 и Cas2, которые присутствуют в подавляющем большинстве известных систем CRISPR-Cas, достаточны для вставки спейсеров в кассеты CRISPR. Эти два белка формируют комплекс, который необходим для процесса адаптации; деятельность эндонуклеазы Cas1 необходима для интеграции спейсера, тогда как Cas2 выполняет неферментативную функцию. Комплекс Cas1-Cas2 представляет собой высококонсервативный модуль "обработки информации" системой CRISPR-Cas, который квазиавтономен от остальной системы. (См. Annotation and Classification of CRISPR-Cas Systems. Makarova KS, Koonin EV. Methods Mol Biol. 2015; 1311:47-75).
[255] Ранее описанные системы класса 2, а именно, типа II и предполагаемого типа V, состояли только из трех или четырех генов в Cas-опероне, а именно, генов cas1 и cas2, составляющих модуль адаптации (пара генов cas1-cas2 не участвует в интерференции), а также единственного многодоменного эффекторного белка, ответственного за интерференцию, но также способствующего процессингу пре-cr-РНК и адаптации, и часто четвертого гена с неохарактеризованными функциями, который необязателен, по меньшей мере, в некоторых системах типа II (и в некоторых случаях четвертый ген - cas4 (данные биохимических или in silico экспериментов показывают, что cas4 принадлежит к суперсемейству нуклеаз PD-(DE)xK с кластером из трех остатков цистеина на C-конце; обладает 5'-экзонуклеазной активностью для оцДНК), или csn2, который кодирует инактивированную АТФ-азу). В большинстве случаев последовательность CRISPR и ген определенной разновидности РНК, известной как tracr-РНК, транс-закодированная небольшая cr-РНК, прилегают к Cas-оперонам класса 2. tracr-РНК частично гомологична повторам в соответствующей последовательности CRISPR и необходима для процессинга пре-cr-РНК, который катализируется РНК-азой III, широко распространенным бактериальным ферментом, не связанным с локусами системы CRISPR-Cas.
[256] Cas1 является самым консервативным белком, который присутствует в большинстве систем CRISPR-Cas и эволюционирует медленнее, чем другие белки Cas. Соответственно, филогения Cas1 использовалась для классификации систем CRISPR-Cas. Результаты биохимических экспериментов или экспериментов in silico показывают, что Cas1 представляет собой металл-зависимую дезоксирибонуклеазу. Делеции в гене Cas1 в геноме E. coli приводят к увеличению чувствительности к повреждению ДНК и ослаблению расхождения хромосом, как описано в "A dual function of the CRISPR-Cassystem in bacterial antivirus immunity and DNA repair," Babu M et al. Mol Microbiol 79:484-502 (2011). Результаты биохимических экспериментов или in silico экспериментов показывают, что Cas2 является РНК-азой, характерной для регионов, насыщенных U, и является двухцепочечной ДНК-азой.
[257] Аспекты изобретения касаются идентификации и конструирования способами инженерии новых эффекторных белков, связанных с системами CRISPR-Cas класса 2. В предпочтительном варианте осуществления эффекторный белок включает односубъединичный эффекторный модуль. В будущем варианте осуществления изобретения эффекторный белок является функциональным в прокариотических или эукариотических клетках для применения in vitro, in vivo или ex vivo. Один аспект изобретения охватывает вычислительные способы и алгоритмы для прогнозирования новых систем CRISPR-Cas класса 2 и определения составляющих их компонентов.
[258] В одном из вариантов осуществления вычислительный способ идентификации новых локусов системы CRISPR-Cas класса 2 включает следующие шаги: обнаружение всех контигов, кодирующих белок Cas1; идентификация всех предсказанных генов, кодирующих белки, в пределах 20 т.п.н. от гена cas1, более конкретно, в пределах 20 т.п.н. от начала гена cas1 и 20 т.п.н. от конца гена cas1; сравнение идентифицированных генов со специфическими профилями белков Cas и предсказание последовательностей CRISPR путем отбора частичных и/или неклассифицированных возможных локусов CRISPR-Cas, содержащих белки с последовательностями длиной более 500 аминокислот (>500 а.к.); анализ отобранных кандидатов с использованием анализов PSI-BLAST и HHPred для выделения и идентификации новых локусов CRISPR-Cas класса 2. В дополнение к вышеупомянутым шагам возможные кандидаты могут быть дополнительно проанализированы путем поиска гомологов по метагеномным базам данных.
[259] Одним из аспектов обнаружения всех контигов, кодирующих белок Cas1, является использование программы GenemarkS для предсказания генов, как более подробно описано в статье "GeneMarkS: a self-training method for prediction of gene starts in microbial genomes. Implications for finding sequence motifs in regulatory regions," John Besemer, Alexandre Lomsadze and Mark Borodovsky, Nucleic Acids Research (2001) 29, pp 2607-2618, включенной в настоящее описание в качестве ссылки.
[260] В одном аспекте проводится идентификация всех предсказанных генов, кодирующих белки, с помощью сравнения идентифицированных генов со специфическими профилями белков Cas и их аннотирования согласно Базе Данных Консервативных Доменов NCBI (CDD), которая состоит из коллекции хорошо аннотированных многочисленных моделей выравнивания последовательностей для древних доменов и полноразмерных белков. Они доступны в виде позиционных весовых матриц (PSSM) для быстрой идентификации консервативных доменов в последовательностях белков посредством анализа RPS-BLAST. CDD включает домены, курируемые NCBI, которые используют информацию о 3D-структуре, чтобы точно определить границы доменов и обеспечить понимание взаимоотношений между последовательностью/структурой/функцией, а также модели домена, импортированные из многих внешних баз данных (Pfam, SMART, COG, PRK, TIGRFAM). В следующем аспекте для предсказания последовательностей CRISPR использовалась программы PILER-CR, которая является общедоступным программным обеспечением для поиска CRISPR-повторов, как описано в статье "PILER-CR: fast and accurate identification of CRISPR repeats", Edgar, R.C., BMC Bioinformatics, Jan 20:8:18(2007), включенной в настоящее описание в качестве ссылки.
[261] В следующем аспекте проводят индивидуальный анализ каждого случая с использованием PSI-BLAST (Средство поиска основного локального выравнивания с учетом позиции). Анализ PSI-BLAST использует позиционную весовую матрицу (PSSM) или профиль множественного выравнивания последовательностей, обнаруженных выше заданного весового порога при белок-белковом анализе BLAST. Данная матрица PSSM используется, чтобы далее искать новые совпадения в базе данных и обновляется для последующих итераций с этими недавно обнаруженными последовательностями. Таким образом, PSI-BLAST является способом обнаружения отдаленных отношений между белками.
[262] В другом аспекте индивидуальный анализ каждого случая предполагает использование HHpred, способа поиска по базам данных последовательностей и предсказания структуры, столь же простого в использовании, как BLAST или PSI-BLAST, и в то же время намного более чувствительного при нахождении отдаленных гомологов. Фактически, чувствительность способа HHpred конкурентоспособна по отношению к самым мощным серверам для предсказания структуры, доступным в настоящее время. HHpred является первым сервером, основанным на попарном сравнении профилей скрытых марковских моделей (СММ). В то время как большинство обычных способов поиска последовательностей выполняют поиск по базам данных последовательностей, таким как UniProt или NR, HHpred выполняет поиск по базам данных выравнивания, таким как Pfam или SMART. Это значительно упрощает получение списка совпадений со многим семействами последовательностей вместо разрозненных единичных последовательностей. Все основные общедоступные базы данных профилей и выравниваний доступны через HHpred. HHpred использует в качестве единичного поискового запроса последовательность или множественное выравнивание. В течение всего нескольких минут HHpred представляет результаты поиска в легком для чтения формате, подобном формату PSI-BLAST. Параметры поиска включают локальное или глобальное выравнивание и весовое сходство вторичной структуры. HHpred может произвести попарные выравнивания последовательностей поискового запроса и шаблона, объединенное множественное выравнивание поискового запроса и шаблона (например, для транзитивного поиска), а также модели 3D-структуры, вычисленные программным обеспечением MODELLER на основе выравниваний HHpred.
[263] Термин "система нацеливания на нуклеиновые кислоты", где нуклеиновая кислота представляет собой ДНК или РНК, и в некоторых аспектах может также относится к гибридам РНК-ДНК или их производным, относится собирательно к транскриптам и другим элементам, участвующим в экспрессии или направлении активности нацеливания на ДНК или РНК генов, ассоциированных с системой CRISPR ("Cas"), которые могут содержать последовательности, кодирующие белки Cas, нацеленные ДНК или РНК, и направляющую РНК, нацеленную ДНК или РНК, включая последовательность cr-РНК (cr-РНК) и (в некоторых, но не всех системах) последовательность РНК, транс-активирующей систему CRISPR/Cas (tracr-РНК), или другие последовательности и транскрипты локуса CRISPR, нацеленные на ДНК или РНК. В целом система нацеливания на РНК характеризуется элементами, способствующими формированию нацеленного на ДНК или РНК комплекса на участке последовательности ДНК или РНК, являющейся мишенью. В контексте образования нацеленного на ДНК или РНК комплекса, термин "последовательность-мишень" относится к последовательности ДНК или РНК, которой комплементарна направляющая РНК, нацеливающая на ДНК или РНК, где гибридизация между последовательностью-мишенью и направляющей РНК, нацеливающей на РНК, способствует образованию нацеливающего на РНК комплекса. В некоторых вариантах осуществления изобретения последовательность-мишень расположена в ядре или цитоплазме клетки.
[264] В одном аспекте изобретения новые системы нацеливания на РНК, также называемые РНК- или РНК-нацеленные CRISPR/Cas или система нацеливания на РНК с помощью системы CRISPR-Cas, описанные в настоящем описании, основаны на идентифицированных белках Cas типа VI, не требующих создания индивидуальных белков для нацеливания на определенные последовательности РНК, кроме единственного фермента, который может быть запрограммирован молекулой РНК для узнавания конкретной ДНК-мишени, иными словами, фермент может быть предназначен для работы с конкретной ДНК-мишенью с помощью указанной молекулы РНК.
[265] В одном аспекте изобретения новые системы нацеливания на ДНК, также называемые ДНК- или ДНК-нацеленные CRISPR/Cas или система нацеливания на ДНК с помощью системы CRISPR-Cas, описанные в настоящем описании, основаны на идентифицированных белках Cas типа VI, не требующих создания индивидуальных белков для нацеливания на конкретные последовательности РНК, кроме единственного фермента, который может быть запрограммирован молекулой РНК для узнавания конкретной ДНК-мишени, иными словами, фермент может быть предназначен для работы с конкретной ДНК-мишени с помощью указанной молекулы РНК.
[266] Системы нацеливания на нуклеиновые кислоты, векторные системы, векторы и композиции, описанные в настоящем описании, могут использоваться в практике нацеливания на нуклеиновые кислоты, изменения или модификация синтеза генных продуктов, таких как белки, расщепления нуклеиновых кислот, редактирования нуклеиновых кислот, сплайсинга нуклеиновых кислот, переноса нуклеиновых кислот-мишеней, отслеживания нуклеиновых кислот-мишеней, выделения нуклеиновых кислот-мишеней, визуализации нуклеиновых кислот-мишеней и т.д.
[267] Термин "белок Cas" или "фермент CRISPR" относится к любому из белков, представленных в новой классификации систем CRISPR-Cas, как описано в настоящем описании.
[268] В предпочтительном варианте осуществления настоящее изобретение охватывает эффекторные белки, относящиеся к типу VI локусов системы CRISPR-Cas, например, локусам C2c2. В настоящем описании C2c2 относится к кандидату 2 класса 2. Локусы C2c2 включают гены cas1 и cas2 наряду с крупным белком, который заявители обозначают как C2c2p, и последовательности CRISPR; однако, C2c2p часто кодируется вблизи последовательностей CRISPR, но не генами cas1-cas2 (для сравнения фиг. 9 и фиг. 15).
Нуклеаза С2с2
[269] Активность C2c2 зависит от присутствия двух доменов HEPN. Показано, что они являются доменами РНК-азы, т.е. нуклеазы (более конкретно, эндонуклеазы), разрезающей РНК. Домен HEPN белка C2c2 также может быть нацелен на ДНК, или потенциально на ДНК и/или РНК. На основании того, что домены HEPN белка C2c2 способны по меньшей мере к связыванию с и, в форме дикого типа, разрезанию РНК, предпочтительно наличие у эффекторного белка C2c2 функции РНК-азы. Этот белок может также, или альтернативно, иметь функцию ДНК-азы.
[270] Таким образом, в некоторых вариантах осуществления эффекторный белок может быть белком, связывающим РНК, таким как белок типа "мертвого"-Cas, который может быть необязательно функционализирован, как описано в настоящем описании, например, активатором транскрипции или доменом репрессора, NLS-последовательностью или другим функциональным доменом. В некоторых вариантах осуществления эффекторный белок может быть РНК-связывающим белком, который расщепляет одиночную цепь РНК. Если связанная РНК является оцРНК, то оцРНК подвергается полному расщеплению. В некоторых вариантах осуществления эффекторный белок может быть РНК-связывающим белком, который расщепляет двойную цепь РНК, например, если он включает два РНК-азных домена. Если связанная РНК является дцРНК, то дцРНК подвергается полному расщеплению.
[271] РНК-азная функция в системах CRISPR известна, например, нацеливание на мРНК было продемонстрировано для определенных систем CRISPR-Cas типа III (Hale et al., 2014, Genes Dev, vol. 28, 2432-2443; Hale et al., 2009, Cell, vol. 139, 945-956; Peng et al., 2015, Nucleic acids research, vol. 43, 406-417), и было показано, что она обеспечивает значительные преимущества. В системе типа III-A Staphylococcus epidermis, транскрипция мишеней приводит к расщеплению ДНК-мишени и ее транскриптов, опосредуемому независимыми активными центрами в эффекторном комплексе рибонуклеопротеина Cas10-Csm (см. Samai et al., 2015, Cell, vol. 151, 1164-1174). Таким образом, изобретение относится к композиции или способу для нацеливания на РНК с помощью эффекторных белков системы CRISPR-Cas.
[272] РНК-мишенью, т.е. представляющей интерес РНК, является РНК, которая на которое нацелено настоящее изобретение, что приводит к привлечению к и связыванию эффекторного белка с представляющим интерес участком-мишенью на РНК-мишени. РНК-мишень может быть любой подходящей формой РНК. В некоторых вариантах осуществления изобретения она может включать мРНК. В других вариантах осуществления изобретения РНК-мишень может включать тРНК или рРНК. В других вариантах осуществления изобретения РНК-мишень может включать микроРНК. В других вариантах осуществления изобретения РНК-мишень может включать миРНК.
Интерферирующая РНК (РНК-i) и микроРНК (мкРНК)
[273] В других вариантах осуществления изобретения РНК-мишень может включать интерферирующую РНК, т.е. РНК, вовлеченную в процесс интерференции РНК, такую как кшРНК, миРНК и т.д. В других вариантах осуществления изобретения РНК-мишень может включать микроРНК (мкРНК). Контроль над интерферирующей РНК или микроРНК может помочь уменьшить неспецифические эффекты (OTE), наблюдаемые в этих подходах, путем уменьшения долговечности интерферирующей РНК или микроРНК in vivo или in vitro.
[274] В определенных вариантах осуществления изобретения мишенью является не сама микроРНК, а микроРНК-связывающий участок микроРНК-мишени.
[275] В определенных вариантах осуществления изобретения микроРНК могут быть изолированы (включая внутриклеточное перемещение). В некоторых вариантах осуществления изобретения микроРНК могут быть разрезаны, например, но не ограничиваясь этим, в шпильках.
[276] В определенных вариантах осуществления изобретения процессинг микроРНК (включая оборот) увеличивается или уменьшается.
[277] Если эффекторный белок и подходящая направляющая молекула селективно экспрессированы (например, под пространственным или временным контролем подходящего промотора, например, промотора, специфичекского для ткани или фазы клеточного цикла, и/или энхансера), это может быть использовано для "защиты" клетки или системы (in vivo или in vitro) от РНК-интерференции в этих клетках. Это может быть полезно для применения в соседних тканях или клетках, где РНК-интерференция не требуется или в целях сравнения с теми клетками или тканями, где эффекторный белок и подходящая направляющая молекула не экспрессируются (т.е. где РНК-интерференция не находится под контролем и где находится, соответственно). Эффекторный белок может быть использован для контроля или связывания с молекулами, включающими или состоящими из РНК, таким как рибозимы, рибосомы или рибопереключатели. В вариантах осуществления изобретения направляющая РНК может нацелить эффекторный белок на все эти молекулы, чтобы эффекторный белок был способен к связыванию с ними.
[278] Система белков изобретения может быть применена в областях технологий РНК-i, без проведения излишних экспериментов, на основе данной заявки, включая терапевтическое, аналитическое и другие применения (см., например, Guidietal, PLoSNeglTropDis 9(5): e0003801. doi: 10.1371/journal.pntd; Crotty et al., InvivoRNAiscreens: conceptsandapplications.Shane Grotty … 2015 Elsevier Ltd. Published by Elsevier Inc., Pesticide Biochemistry and Physiology (Impact Factor: 2.01). 01/2015; 120. DOI: 10.1016/j.pestbp.2015.01.002 and Makkonen et al., Viruses 2015, 7(4), 2099-2125; doi:10.3390/v7042099), потому что настоящая заявка обеспечивает основу для осознанной модификации системы.
Рибосомная РНК (рРНК)
[279] Азалиды, такие как азитромицин, являются широко известными антибиотиками. Они нацелены на и разрушают рибосомную субъединицу 50S. В некоторых вариантах осуществления изобретения данный эффекторный белок, вместе с подходящей направляющей РНК для нацеливания на рибосомную субъединицу 50S может быть нацелен на связывание с рибосомной субъединицей 50S. Таким образом, изобретение относится к эффекторному белку по настоящему изобретению совместно с подходящей направляющей молекулой, направленной на рибосомную (особенно рибосомную субъединицу 50S) мишень. Использование этого эффекторного белка совместно с подходящей направляющей молекулой, направленной на рибосомную (в особенности субъединицу 50S) мишень, может быть использовано в качестве антибиотика. В частности, действие в качестве антибиотика аналогично действию азалидов, таких как азитромицин. В некоторых вариантах осуществления изобретения мишенью могут быть прокариотические рибосомные субъединицы, такие как субъединица 70S у прокариот, вышеупомянутая субъединица 50S, субъединица 30S, а также 16S и 5S субъединицы. В других вариантах осуществления изобретения мишенью могут быть эукариотические рибосомные субъединицы, такие как субъединица 80S у эукариот, субъединица 60S, субъединица 40S, а также субъединицы 28S, 18S, 5,8S и 5S.
[280] В некоторых вариантах осуществления изобретения эффекторный белок может быть РНК-связывающим белком, необязательно функционализированным, как описано в настоящем описании. В некоторых вариантах осуществления изобретения эффекторный белок может быть РНК-связывающим белком, расщепляющим одиночную цепь РНК. Во всех случаях, но особенно когда РНК-связывающий белок расщепляет одиночную цепь РНК, рибосомная функция может быть модулирована и, в частности, уменьшена или разрушена. Это может относиться к любой рибосомной РНК и любой рибосомной субъединице, так как последовательности рРНК хорошо известны.
[281] Таким образом, предусматривается контроль рибосомной активности посредством использования данного эффекторного белка совместно с подходящей направляющей молекулой для рибосомной мишени. Контроль может осуществляться посредством расщепления или связывания с рибосомой. В частности, предусматривается сокращение активности рибосомы. Это может быть полезно для анализа функций рибосом in vivo или in vitro, но также и как средство контроля в терапевтических способах, основанных на рибосомной активности, in vivo или in vitro. Кроме того, предусматривается контроль (т.е. сокращение) синтеза белка в системах in vivo или in vitro, такой контроль может быть полезен при использовании в качестве антибиотика, а также в исследовательских и диагностических целях.
Рибопереключатели
[282] Рибопереключатель (также известный как аптозим) является регуляторным сегментом молекулы матричной РНК, которая связывает низкомолекулярное соединение. Это связывание, как правило, приводит к изменению синтеза белков, закодированных мРНК. Таким образом, предусматривается контроль активности рибопереключателя посредством использования данного эффекторного белка совместно с подходящей направляющей молекулой для рибопереключателя-мишени. Контроль может осуществляться путем расщепления или связывания с рибопереключателем. В частности, предусматривается сокращение активности рибопереключателя. Это может быть полезно при анализе функций рибопереключателя in vivo или in vitro, но также и как средство контроля терапевтических способах, основанных на активности рибопереключателя, in vivo или in vitro. Кроме того, предусматривается контроль (т.е. сокращение) синтеза белка в системах in vivo или in vitro. Что касается рРНК, этот контроль может быть полезен при использовании в качестве антибиотика, а также в исследовательских и диагностических целях.
Рибозимы
[283] Рибозимы являются молекулами РНК, имеющими каталитические свойства, аналогичные ферментам (которые являются белками). Поскольку рибозимы, как естественные, так и искусственно спроектированные, включают или полностью состоят из РНК, они также могут быть мишенью РНК-связывающего эффекторного белка по настоящему изобретению. В некоторых вариантах осуществления изобретения эффекторный белок может быть РНК-связывающим белком, расщепляющим рибозимы для их отключения. Так, контроль рибозимной активности предусматривается посредством использования эффекторного белка по настоящему изобретению совместно с подходящей направляющей молекулой для рибозима-мишени. Контроль может осуществляться путем расщепления или связывания с рибозимом. В частности, предусматривается сокращение рибозимной активности. Это может быть полезно при анализе функции рибозим in vivo или in vitro, но также и как средство контроля в способах терапии на основе рибозимной активности, in vivo или in vitro.
Экспрессиягенов, включая процессинг РНК
[284] Эффекторный белок может также использоваться вместе с подходящей направляющей молекулой для нацеливания на экспрессию генов, в том числе посредством контроля процессинга РНК. Контроль процессинга РНК может включать реакции процессинга РНК, такие как сплайсинг РНК, включая альтернативный сплайсинг, путем нацеливания на РНК-полимеразы; вирусную репликацию (в особенности, вирусов-сателлитов, бактериофагов и ретровирусов, таких как вирус гепатита B (HBV), вирус гепатита С (HCV) и ВИЧ и другие, перечисленные в настоящем описании) включая вироиды в растениях; и биосинтез тРНК. Эффекторный белок и подходящая направляющая молекула могут также использоваться для контроля активации РНК (РНК-a). Активация РНК стимулирует экспрессию генов, таким образом, контроль экспрессии генов может быть достигнут посредством нарушения или сокращения активации РНК и таким образом меньшей стимуляции экспрессии генов. Механизмы осуществления контроля подробнее обсуждаются ниже.
Скрининг РНК-интерференции
[285] Генные продукты, нокдаун которых связан с изменениями фенотипа и биологическими каскадами, могут быть проанализированы, а их составные части идентифицированы путем скрининга РНК-i. Контроль может осуществляться во время скрининга при помощи эффекторного белка и подходящей направляющей молекулы, чтобы прекратить РНК-i или уменьшить ее активность во время скрининга и таким образом восстановить активность (ранее участвовавшего в РНК-i) генного продукта (прекращая или уменьшая интерференцию/репрессию).
[286] Сателлитные РНК (sat-РНК) и вирусы-сателлиты могут также быть преобразованы.
[287] Контроль в данном случае осуществляется посредством РНК-азной активности, что обычно означает сокращение, нарушение, нокдаун или нокаут данной активности.
Способы применения РНК in vivo
Ингибирование экспрессии генов
[288] Мишень-специфические РНК-азы, описанные в настоящем описании, позволяют очень точно разрезать РНК-мишень. Интерференция на уровне РНК обеспечивает модулирование в пространстве и времени неинвазивным способом, поскольку не вносит изменений в геном.
[289] Было продемонстрировано, что многие болезни поддаются лечению с помощью нацеливания на мРНК. В то время как большинство этих исследований касается применения малой интерферирующей РНК, ясно, что РНК, нацеливающая эффекторные белки, описанные в настоящем описании, может быть применена сходным образом.
[290] Примерами мРНК-мишеней (и соответствующих способов лечения заболеваний) является VEGF, VEGF-R1 и RTP801 (в лечении старческой дегенерации желтого пятна (AMD) и/или диабетического макулярного отека (DME)), каспаза 2 (в лечении неартериитической передней ишемической невропатии зрительного нерва (NAION)), ADRB2 (в лечении внутриглазного давления), TRFVI (в лечении синдрома сухого глаза), киназа Syk (в лечении астмы), Apo В (в лечении гиперхолестеринемии), PLK1, KSP и VEGF (в лечении солидных опухолей), Ber-Ab1 (в лечении хронического миелоидного лейкоза (CML)) (Burnett and Rossi Chem Biol. 2012, 19(1): 60-71). Точно также была продемонстрирована эффективность нацеливания на РНК при лечении заболеваний, вызываемых РНК-вирусами, такими как ВИЧ (HIV) (нацеливание на Tet и Rev HIV), респираторно-синцитиального вируса (RSV) (нацеливание на нуклеокапсид RSV) и вируса гепатита C (HCV) (нацеливание на miR-122) (Burnett and Rossi Chem Biol. 2012, 19(1): 60-71).
[291] Кроме того, предусматривается, что нацеливание на РНК эффекторного белка по изобретению может использоваться для прицельного нокдауна как определенной мутации так и целого аллеля. Направляющие РНК могут быть разработаны для специфичного нацеливания на последовательность в транскрибированной мРНК, содержащей мутацию или последовательность определенного аллеля. Такой специфический нокдаун особенно подходит для терапевтического применения в случае заболеваний, связанных с генными продуктами, содержащими мутации или определенный аллель. Например, большинство случаев семейной гипобеталипопротеинемии (FHBL) вызвано мутациями в гене ApoB. Этот ген кодирует две версии белка аполипопротеина В: короткую версию (ApoB-48) и более длинную версию (ApoB-100). Несколько мутаций в гене ApoB, которые приводят к FHBL, приводят к тому, что обе версии белка ApoB являются аномально короткими. Специфическое нацеливание на и нокдаун транскриптов мРНК, содержащих мутантный ген ApoB, с помощью РНК-нацеленного эффекторного белка по изобретению, может быть полезен для лечения FHBL. Другим примером может служить болезнь Хантингтона (HD), которую вызывает распространение повторов триплета CAG в гене, кодирующем белок хантингтин, что приводит к производству аномального белка. Специфическое нацеливание на и нокдаун транскриптов мРНК, содержащих мутации или определенный аллель, кодирующий аномальный белок хантингтин, с помощью РНК-нацеленного эффекторного белка по изобретению, может быть полезным для лечения болезни Хантингтона (HD).
[292] Отмечено, что в этом контексте, и более широко для различных способов применения, как описано в настоящем описании, может быть предусмотрено использование разделенной версии РНК-нацеленного эффекторного белка. Действительно, эта версия может не только обеспечить высокую точность, но и быть более удобной для доставки. C2c2 разделен в том смысле, что две части фермента C2c2, по существу, составляют функциональный C2c2. В идеале, разделение не должно затрагивать каталитический домен(ы) С2с2. Этот C2c2 может функционировать как нуклеаза или это может быть "мертвый"-C2c2, который по сути является РНК-связывающим белком с очень небольшой каталитической активностью или полным ее отсутствием, как правило, в результате мутации (мутаций) в каталитических доменах.
[293] Каждая половина разделенного C2c2 может быть слита с партнером по димеризации. В качестве неограничивающего примера, использование доменов димеризации, чувствительных к рапамицину, позволяет производить химически индуцируемое разделение C2c2 для временного контроля активности C2c2. Таким образом, C2c2 может быть химически индуцируемым, будучи разделенным на два фрагмента, и для контролируемой повторной сборки C2c2 могут использоваться чувствительные к рапамицину домены димеризации. Две части разделенного C2c2 могут считаться N'-концевой частью и C'-концевой частью разделенного C2c2. Соединение, как правило, осуществляется в точке разделения C2c2. Иными словами, С'-концевая часть N'-концевой части разделенного C2c2 соединена с одной из половин димера, в то время как N'-концевая часть C'-концевой части соединена с другой половиной димера.
[294] C2c2 не должен быть разделен в том смысле, что разрыв образуется заново. Точка разделения, как правило, определяется in silico и клонируется в конструкции. Вместе две части разделенного C2c2, N'-концевые и C'-концевые части, формируют целый C2c2, включающий предпочтительно по меньшей мере 70% или более аминокислот дикого типа (или нуклеотидов, их кодирующих), предпочтительно по меньшей мере 80% или более, предпочтительно по меньшей мере 90% или более, предпочтительно по меньшей мере 95% или более, и наиболее предпочтительно по меньшей мере 99% или более аминокислот дикого типа (или нуклеотидов, их кодирующих). Некоторые модификации возможны, и предусматриваются мутанты. Нефункциональные домены могут быть полностью удалены. Важно, что в результате объединения этих двух частей желаемая функция C2c2 может быть восстановлена или воссоздана. Димер может быть гомодимером или гетеродимером.
[295] В некоторых вариантах осуществления изобретения эффекторный белок C2c2, как описано в настоящем описании, может использоваться для нацеливания, специфического для конкретной мутации или аллеля, такого как нокдаун гена, специфический для мутации или аллеля.
[296] РНК-нацеленный эффекторный белок, кроме того, может быть соединен с другим функциональным РНК-азным доменом, таким как неспецифичная РНК-аза или Argonaute 2, для совместного увеличения РНК-азной активности или гарантированной дальнейшей деградации РНК.
[297] Модулирование экспрессии генов посредством модуляции функции РНК
[298] Кроме прямого влияния на экспрессию генов посредством расщепления мРНК, нацеливание на РНК может также использоваться для влияния на конкретные аспекты процессинга РНК в клетке, что может позволить более тонкое модулирование экспрессии генов. В основном модулирование может быть опосредовано, например, вмешательством в связывание белков с РНК, как, например, при блокировании связывания белков или привлечении РНК-связывающих белков. Действительно, модулирование может быть обеспечено на разных уровнях, таких как сплайсинг, транспорт, локализация, трансляция и обновление мРНК. Также в контексте терапии, может быть предусмотрена адресное воздействие на (патогенные) сбои на каждом из этих уровней при помощи РНК-специфичных нацеленных молекул. В этих вариантах осуществления изобретения во многих случаях предпочтительно использование "мертвого" C2c2, который потерял способность разрезать РНК-мишень, но способен к связыванию с ней, такого как мутантные формы C2c2, описанного в настоящем описании, в качестве белка для нацеливания на РНК.
a) альтернативный сплайсинг
[299] Многие гены человека имеют многочисленные мРНК, образованные в результате альтернативного сплайсинга. Показано, что различные болезни связаны с отклонениями сплайсинга, приводящими к потере или приобретению функции экспрессируемым геном. В то время как некоторые из этих болезней вызваны мутациями, которые вызывают дефекты сплайсинга, но много из них - нет. Один вариант терапии состоит в нацеливании непосредственно на механизм сплайсинга. РНК, нацеливающая эффекторные белки, описанные в настоящем описании, может использоваться, например, для блокирования или стимуляции разрезания, включения или исключения экзонов, влияния на экспрессию специфичных изоформ и/или стимуляции экспрессии альтернативных продуктов белка. Такие способы применения более подробно описаны ниже.
[300] Эффекторный белок нацеливания на РНК при связывании с РНК-мишенью может пространственно блокировать доступ факторов сплайсинга к последовательности РНК. Эффекторный белок нацеливания на РНК, нацеленный на участок сплайсинга, может блокировать сплайсинг в этом участке, необязательно перенаправляя сплайсинг к соседнему участку. Например, эффекторный белок нацеливания на РНК при связывании с 5'-концом сайта сплайсинга, может блокировать связывание с компонентом U1 сплайсосомы, что приводит к пропуску этого экзона. Альтернативно, эффекторный белок нацеливания на РНК, нацеливаясь на энхансер или сайленсер сплайсинга, может предотвратить связывание трансактивирующих регуляторных факторов сплайсинга в участке-мишени и эффективно блокировать или стимулировать сплайсинг. Исключение экзона может далее быть достигнуто связыванием ILF2/3 с предшественником мРНК около экзона с помощью эффекторного белка нацеливания на РНК, как описано в настоящем описании. В качестве другого примера, может быть присоединен богатый глицином домен для связывания с hnRNP A1 и исключения экзона (Del Gatto-Konczak et al. Mol Cell Biol. 1999 Jan; 19(1):251-60).
[301] В некоторых вариантах осуществления изобретения посредством соответствующего подбора направляющей РНК, мишенью могут быть конкретные варианты сплайсинга, в то время как другие варианты сплайсинга могут не быть мишенью.
[302] В некоторых случаях эффекторный белок нацеливания на РНК может использоваться, чтобы способствовать сплайсингу (например, в участках, где сплайсинг проходит с нарушениями). Например, эффекторный белок нацеливания на РНК может быть связан с эффектором, способным к стабилизации регуляторной структуры например шпильки, для продолжения сплайсинга. Эффекторный белок нацеливания на РНК может быть связан с консенсусной последовательностью участка связывания для конкретного фактора сплайсинга, чтобы обеспечить связывание белка с ДНК-мишенью.
[303] Примеры заболеваний, связанных с отклонениями сплайсинга, включают, но не ограничиваются ими, паранеопластическую опсоклонус-миоклонус атаксию (POMA), возникающей при утрате белков Nova, регулирующих сплайсинг белков, которые функционируют в синапсах, и муковисцидоз, который вызван нарушенным сплайсингом регулятора трансмембранной проводимости муковисцидоза, приводящим к потере нефункциональности хлоридными каналами. При других заболеваниях отклонения сплайсинга РНК приводят к мутации приобретения функции. Например, в случае миотонической дистрофии, которая вызвана распространением повторов триплета CUG (от 50 до> 1500 повторов) в 3'-нетранслируемой области (3'НТО) мРНК, что вызывает дефекты сплайсинга.
[304] Эффекторный белок нацеливания на РНК может использоваться для включения экзона посредством присоединения фактора сплайсинга (таким как U1) к 5'-сайту сплайсинга, что способствует вырезанию интронов вокруг желаемого экзона. Такое связывание может быть опосредовано путем присоединения домена, богатого аргинином/серином, который функционирует как активатор сплайсинга (Gravely BR and Maniatis T, Mol Cell. 1998 (5):765-71).
[305] Предусматривается, что эффекторный белок нацеливания на РНК может использоваться, чтобы заблокировать механизм сплайсинга в желаемом локусе для предотвращения распознавания экзона и экспрессии другого белка. Примером заболевания, которое может быть вылечено этим способом, является миодистрофия Дюшенна (DMD), которая вызвана мутациями в гене, кодирующем белок дистрофин. Почти все мутации, ведущие к миодистрофии Дюшенна (DMD), являются мутациям сдвига рамки считывания, приводящими к ухудшению трансляции дистрофина. Эффекторный белок нацеливания на РНК может быть соединен с участками сшивки или экзонными энхансерами сплайсинга (ESE), таким образом, предотвращая распознавание экзона, что приводит к трансляции частично функционального белка. Это преобразовывает фенотип Дюшенна, приводящий к смерти, в более мягкий фенотип Беккера с менее серьезными симптомами.
b) Модификация РНК
[306] Редактирование РНК - естественный процесс, посредством которого разнообразие генных продуктов данной последовательности увеличивается в результате незначительной модификации РНК. Как правило, модификация включает преобразование аденозина (A) в инозин (I), что приводит к формированию последовательности РНК, отличающейся от закодированной в геноме. Модификацию РНК обычно производит фермент ADAR, в ходе модификации пре-РНК-мишень формирует несовершенную дуплексную РНК путем спаривания оснований последовательности экзона, который содержит аденозин, нуждающийся в редактировании, и интронного некодирующего элемента. Классическим примером редактирования аденозина (A) в инозин (I) является мРНК рецептора глутамата GluR-B, где редактирование приводит к изменению свойств проводимости канала (Higuchi M, et al.Cell. 1993; 75:1361-70).
[307] У людей гетерозиготная функционально-нулевая мутация в гене ADAR1 приводит к кожному заболеванию, пигментному генодерматозу человека (Miyamura Y, et al.Am J Hum Genet. 2003; 73:693-9). Предусматривается, что эффекторные белки, нацеленные на РНК, по настоящему изобретению могут быть использованы, чтобы исправить сбои в модификации РНК.
[308] Далее предусматривается, что аденозиновая метилаза РНК (N (6)-метиладенозин) может быть присоединена к эффекторным белкам нацеливания на РНК по изобретению и нацелена на представляющий интерес транскрипт. Эта метилаза вызывает обратимое метилирование, выполняет регуляторную роль и может влиять на экспрессию генов и решение судьбы клетки, модулируя многие клеточные процессы, связанные с РНК. (Fu et al Nat Rev Genet. 2014; 15(5):293-306).
c) Полиаденилирование
[309] Полиаденилирование мРНК важно для транспорта в ядро, эффективности трансляции и стабильности мРНК; все эти процессы, как и процесс полиаденилирования, зависят от определенных РНК-связывающих белков. Большинство эукариотических мРНК после транскрипции получают 3'-поли(А)-хвостовую часть приблизительно из 200 нуклеотидов. В полиаденилировании участвуют различные РНК-связывающие белковые комплексы, стимулирующие активность поли(А)-полимеразы (Minvielle-Sebastia L et al. Curr Opin Cell Biol. 1999; 11:352-7). Предусматривается, что эффекторные белки нацеливания на РНК, описанные в настоящем описании, могут быть использованы для вмешательства в или стимуляции взаимодействия между РНК-связывающими белками и РНК.
[310] Примером заболевания, связанного с дефектными белками, участвующими в полиаденилировании, является окулофарингеальная мышечная дистрофия (OPMD) (Brais B, et al. Nat Genet. 1998,18:164-7).
d) Экспорт РНК
[311] После процессинга пре-мРНК, мРНК экспортируется из ядра в цитоплазму. Это обеспечивается клеточным механизмом, который включает образование комплекса белка-переносчика и мРНК, который потом перемещается через ядерную пору и высвобождает мРНК в цитоплазме с последующей переработкой переносчика.
[312] На Xenopus показано, что избыточная экспрессия белков (таких как TAP), играющих роль в экспорте РНК, увеличивает экспорт транскриптов, которые иначе экспортируются неэффективно (Katahira J, et al.EMBO J. 1999; 18:2593-609).
e) Локализация мРНК
[313] Локализация мРНК необходима для пространственного регулирования синтеза белка. Локализация транскриптов в определенных компартментах клетки может быть обеспечена сигналами локализации. В конкретных вариантах осуществления изобретения предусматривается, что эффекторные белки, описанные в настоящем описании, могут использоваться для нацеливания на сигналы локализации в представляющих интерес РНК. Эффекторные белки могут быть сконструированы для связывания транскрипта-мишени и его доставки в определенный клеточный компартмент согласно его сигнальной последовательности. В частности, для изменения локализации РНК может использоваться, например, эффекторный белок нацеливания на РНК слитый с сигналом ядерной локализации (NLS).
[314] Другие примеры сигналов локализации включают связывающий zip-код белок (ZBP1), который обеспечивает локализацию β-актина в цитоплазме некоторых асимметричных типов клеток, последовательность KDEL (локализация в эндоплазматическом ретикулуме), сигнал ядерного экспорта (NES) (локализация в цитоплазме), сигнал нацеливания в митохондрим (локализация в митохондриях), сигнал нацеливания в пероксисомы (локализация в пероксисоме) и m6A-метка/YTHDF2 (локализация в p-телах). Другие подходы, которые предусматриваются, включают слияние эффекторного белка нацеливания на РНК с белками известной локализации (например, мембрана, синапс).
[315] Альтернативно, эффекторный белок согласно изобретению может использоваться, например, в зависимом от локализации нокдауне. Путем слияния эффекторного белка с соответствующим сигналом локализации эффектор может быть нацелен на определенный клеточный компартмент. Эффективное нацеливание будет осуществлено только для РНК-мишеней, находящихся в этом компартменте, тогда как нацеливание на другие мишени, во всех отношениях идентичные, но находящиеся в другом клеточном компартменте, не будет осуществлено, таким образом может быть организован нокдаун, зависящий от локализации.
f) трансляция
[316] Эффекторные белки нацеливания на РНК, описанные в настоящем описании, могут использоваться для усиления или подавления трансляции. Предусматривается, что регуляция трансляции является очень мощным способом управления клеточными процессами. Кроме того, для функциональных исследований скрининг трансляции белка может быть более полезным по сравнению со скринингом повышенной транскрипции, имеющим тот недостаток, что увеличение числа транскриптов не приводит к повышению синтеза белка.
[317] Предусматривается, что эффекторные белки нацеливания на РНК, описанные в настоящем описании, могут использоваться для переноса факторов инициации трансляции, таких как EIF4G около 5'-нетранслируемой области (5'-НТО) матричной РНК-мишени для управления трансляцией (как описано в De Gregorio et al.EMBO J. 1999; 18(17):4865-74 для неперепрограммируемого РНК-связывающего белка). В качестве другого примера, GLD2, цитоплазматическая поли(А)-полимераза, может быть привлечена к мРНК-мишени эффекторным белком нацеливания на РНК. Таким образом, можно осуществлять направленное полиаденилирование мРНК-мишени в целях стимуляции трансляции.
[318] Точно так же эффекторные белки нацеливания на РНК, предусматриваемые в рамках настоящего изобретения, могут использоваться для блокирования репрессоров трансляции мРНК, таких как ZBP1 (Huttelmaier S, et al.Nature. 2005; 438:512-5). Непосредственная регуляция трансляции может быть достигнута путем связывания с участком инициации трансляции РНК-мишени.
[319] Кроме того, с помощью слияния эффекторных белков нацеливания на РНК с белком, стабилизирующим мРНК, например, предотвращающим его деградацию, таким как ингибиторы РНК-азы, можно увеличить синтез белка с представляющих интерес транскриптов.
[320] Предусматривается, что эффекторные белки нацеливания на РНК, описанные в настоящем описании, могут использоваться для подавления трансляции посредством связывания с 5'-нетранслируемыми областями (5'-НТО) транскрипта РНК, что препятствует сборке рибосомы и началу трансляции.
[321] Далее, эффекторный белок нацеливания на РНК может использоваться для связывания Caf1, компонента комплекса деаденилазы CCR4-NOT, с мРНК-мишенью, что приводит к деаденилированию транскрипта-мишени и ингибированию трансляции белка.
[322] Например, эффекторный белок нацеливания на РНК по изобретению может использоваться, чтобы увеличить или уменьшить трансляцию белков, представляющих терапевтический интерес. Примерами терапевтического применения, где эффекторный белок нацеливания на РНК может использоваться для понижения или повышения трансляции, могут служить боковой амиотрофический склероз (ALS) и сердечно-сосудистые заболевания. При ALS показано снижение уровня глиального транспортера глутамата EAAT2 в двигательной зоне коры головного мозга и спинном мозге, а также множественные аномальные транскрипты мРНК EAAT2 в тканях головного мозга. Считается, что отсутствие белка EAAT2 является главной причиной эксайтотоксичности при ALS. Восстановление уровня белка EAAT2 и его функции может дать терапевтический эффект. Следовательно, эффекторный белок нацеливания на РНК может быть использован для повышения экспрессии белка EAAT2, например, посредством блокирования репрессоров трансляции или стабилизации мРНК, как описано выше. Аполипопротеин A1 (ApoA1) является главным белковым компонентом липопротеинов высокой плотности (HDL), как ApoA1, так и HDL, обычно рассматривают как атеропротекторы (снижают риск развития атеросклероза). Предусматривается, что эффекторный белок нацеливания на РНК может быть использован для повышения экспрессии ApoA1, например, путем блокирования репрессоров трансляции или стабилизации мРНК, как описано выше.
g) Обмен мРНК
[323] Трансляция тесно связана с обменом мРНК и регулируемой стабильностью мРНК. Показано, что конкретные белки поддерживают стабильность транскриптов, такие как белки ELAV/Hu в нейронах (Keene JD, 1999, Proc Natl Acad Sci U S A. 96:5-7) и тристетрапролин (TTP). Эти белки стабилизируют мРНК-мишень, защищая их от деградации в цитоплазме (Peng SS et al., 1988, EMBO J. 17:3461-70).
[324] Предусматривается, что эффекторные белки нацеливания на РНК по настоящему изобретению могут использоваться для вмешательства или стимулирования активности белков, стабилизирующих транскрипты мРНК, и таким образом влиять на обновление мРНК. Например, связывание TTP человека с РНК-мишени с использованием эффекторного белка нацеливания на РНК может приводить к опосредуемой аденилат-уридилат-богатым элементом (AU-богатым элементом) репрессии трансляции и деградации мишени. AU-богатые элементы, способствующие стабильности РНК, найдены в 3'-нетранслируемой области (3'-НТО) многих мРНК, кодирующих протоонкогены, ядерные транскрипционные факторы и цитокины. В качестве другого примера, эффекторный белок нацеливания на РНК может быть слит с HuR, другим белком стабилизации мРНК (Hinman MN and Lou H, Cell Mol Life Sci 2008; 65:3168-81), чтобы присоединить его к транскрипту-мишени для увеличения времени жизни этого транскрипта или стабилизации короткоживущей мРНК.
[325] Далее предусматривается, что эффекторные белки нацеливания на РНК, описанные в настоящем описании, могут использоваться для деградации транскриптов-мишеней. Например, m6A-метилтрансфераза может быть присоединена к транскрипту-мишени, чтобы транспортировать транскрипт к P-телам с целью его последующей деградации.
[326] В качестве другого примера, эффекторный белок нацеливания на РНК, как описано в настоящем описании, может быть слит с неспецифичным эндонуклеазным доменом Pilt N-конца (PIN), чтобы присоединить его к транскрипту-мишени и обеспечить последующую деградацию этого транскрипта.
[327] У пациентов, страдающих энцефаломиелитом и невропатией, связанными с паранеопластическим неврологическим расстройством (PND), вырабатываются аутоантитела против Hu-белков в опухолях за пределами центральной нервной системы (Szabo et al. 1991, Клетка.; 67:325-33), которые потом пересекают гематоэнцефалический барьер. Предусматривается, что эффекторные белки нацеливания на РНК по настоящему изобретению могут использоваться, чтобы препятствовать связыванию аутоантител с транскриптами мРНК.
[328] При миотонической дистрофии типа 1 (DM1), вызванной распространением повторов (CUG)n в 3'-нетранслируемой области (3'-НТО) гена протеинкиназы мышечной дистрофии (DMPK) происходит накопление аномальных транскриптов в ядре. Предусматривается, что эффекторные белки нацеливания на РНК по изобретению, слитые с эндонуклеазой для нацеливания на повторы (CUG)n, могут препятствовать накоплению таких транскриптов.
h) Взаимодействие с многофункциональными белками
[329] Некоторые РНК-связывающие белки связываются со многими участкам на многочисленных РНК, чтобы принимать участие в разнообразных процессах. Например, показано, что белок hnRNP A1 связывает экзонные последовательности сайленсера сплайсинга, противодействуя факторам сплайсинга, ассоциированными с концами теломер (таким образом, стимулируя активность теломер), а также связывает микроРНК, чтобы облегчить Drosha-опосредованный процессинг, таким образом, влияя на созревание. Предусматривается, что РНК-связывающие эффекторные белки по настоящему изобретению изобретения могут вмешиваться в связывание РНК-связывающих белков c РНК в одном или более местоположениях.
i) Фолдинг РНК
[330] РНК принимает определенную структуру для выполнения своих биологических функций. Изменения конформации между альтернативными третичными структурами очень важны для большинства РНК-опосредованных процессов. Однако проблема фолдинга РНК имеет несколько составляющих. Например, РНК может иметь тенденцию сворачиваться и поддерживать неподходящую альтернативную конформацию и/или правильная третичная структура может быть недостаточно термодинамически выгодной по сравнению с альтернативными структурами. Эффекторный белок нацеливания на РНК по изобретению, в особенности, не имеющий способности к расщеплению, или "мертвый" белок нацеливания на РНК, может использоваться непосредственно для фолдинга (м)РНК и/или поддержания ее правильной третичной структуры.
Использование эффекторного белка нацеливания на РНК для изменения состояния клетки
[331] В определенных вариантах осуществления изобретения C2c2 в комплексе с cr-РНК активируется после связывания с РНК-мишенью и впоследствии расщепляет любые соседние оцРНК-мишени (т.е. "побочный эффект" или "эффект свидетеля"). C2c2, однажды активированный собственной мишенью, может расщепить другие (некомплементарные) молекулы РНК. Такое неспецифичное расщепление РНК может потенциально обладать цитотоксическим действием, или иным образом влиять на клеточную физиологию или состояние клетки.
[332] Соответственно, в некоторых вариантах осуществления изобретения, не встречающаяся в природе или сконструированная способами инженерии композиция, векторная система или система доставки, как описано в настоящем описании, используются или могут использоваться для индукции состояния покоя клеток. В некоторых вариантах осуществления изобретения не встречающаяся в природе или сконструированная способами инженерии композиция, векторная система или система доставки, как описано в настоящем описании, используются или могут использоваться для индукции остановки клеточного цикла. В некоторых вариантах осуществления изобретения не встречающаяся в природе или сконструированная способами инженерии композиция, векторная система или система доставки, как описано в настоящем описании, используются или могут использоваться для подавления роста и/или пролиферации клеток. В некоторых вариантах осуществления изобретения не встречающаяся в природе или сконструированная способами инженерии композиция, векторная система или система доставки, как описано в настоящем описании, используются или могут использоваться для индукции анергии клетки. В некоторых вариантах осуществления изобретения не встречающаяся в природе или сконструированная способами инженерии композиция, векторная система или система доставки, как описано в настоящем описании, используются или могут использоваться для индукции апоптоза клетки. В некоторых вариантах осуществления изобретения не встречающаяся в природе или сконструированная способами инженерии композиция, векторная система или система доставки, как описано в настоящем описании, используются или могут использоваться для индукции некроза клетки. В некоторых вариантах осуществления изобретения не встречающаяся в природе или сконструированная способами инженерии композиция, векторная система или система доставки, как описано в настоящем описании, используются или могут использоваться для индукции клеточной гибели. В некоторых вариантах осуществления изобретения не встречающаяся в природе или сконструированная способами инженерии композиция, векторная система или система доставки, как описано в настоящем описании, используются или могут использоваться для индукции программируемой клеточной гибели.
[333] В некоторых вариантах осуществления изобретение касается способа индукции состояния покоя клетки путем введения не встречающейся в природе или сконструированной способами инженерии композиции, векторной системы или системы доставки, как описано в настоящем описании. В некоторых вариантах осуществления изобретение относится к способу индукции остановки клеточного цикла путем введения не встречающейся в природе или сконструированной способами инженерии композиции, векторной системы или системы доставки, как описано в настоящем описании. В некоторых вариантах осуществления изобретение касается способа подавления роста и/или пролиферации клеток путем введения не встречающейся в природе или сконструированной способами инженерии композиции, векторной системы или системы доставки, как описано в настоящем описании. В некоторых вариантах осуществления изобретение касается способа для индукции анергии клетки путем введения не встречающейся в природе или сконструированной способами инженерии композиции, векторной системы или системы доставки, как описано в настоящем описании. В некоторых вариантах осуществления изобретение касается способа индукции апоптоза клетки путем введения не встречающейся в природе или сконструированной способами инженерии композиции, векторной системы или системы доставки, описанной в настоящей заявке. В некоторых вариантах осуществления изобретения изобретение касается способа для индукции некроза клетки путем введения не встречающейся в природе или сконструированной способами инженерии композиции, векторной системы или системы доставки, как описано в настоящем описании. В некоторых вариантах осуществления изобретение касается способа для индукции клеточной гибели путем введения не встречающейся в природе или сконструированной способами инженерии композиции, векторной системы или системы доставки, как описано в настоящем описании. В некоторых вариантах осуществления изобретения изобретение касается способа для индукции программируемой клеточной гибели путем введения не встречающейся в природе или сконструированной способами инженерии композиции, векторной системы или системы доставки, как описано в настоящем описании.
[334] Способы и применения, как описано в настоящем описании, могут быть терапевтическими или профилактическими и могут быть нацелены на отдельные клетки, клеточные (суб)популяции или типы клеток/тканей. В частности, способы и типы использования, как описано в настоящем описании, могут быть терапевтическими или профилактическими и могут быть нацелены на отдельные клетки, клеточные (суб)популяции или типы клеток/тканей, экспрессирующие одну или более последовательностей-мишеней, таких как один или более определенных РНК-мишеней (например, оцРНК). Без ограничения, клетки-мишени могут быть, например, раковыми клетками, экспрессирующими определенный транскрипт, например, нейроны определенного класса, (иммунные) клетки, вызывающие, например, аутоиммуные процессы, или клетки, зараженные конкретным (например, вирусным) патогеном, и т.д.
[335] Соответственно, в некоторых вариантах осуществления изобретение относится к способам лечения патологических состояний, характеризующихся присутствием нежелательных клеток (клеток-хозяев), включающим введение или индукцию не встречающихся в природе или сконструированных способами инженерии композиций, векторных систем или систем доставки, как описано в настоящем описании. В некоторых вариантах осуществления изобретение относится к применению не встречающихся в природе или сконструированных способами инженерии композиций, векторных систем или систем доставки, как описано в настоящем описании, для лечения патологических состояний, характеризующихся присутствием нежелательных клеток (клеток-хозяев). В некоторых вариантах осуществления изобретение относится к не встречающимися в природе или сконструированным способами инженерии композициям, векторным системам или системам доставки, как описано в настоящем описании, для применения в лечении патологических состояний, характеризующихся присутствием нежелательных клеток (клеток-хозяев). Необходимо понимать, что система CRISPR-Cas предпочтительно нацелена на мишени, специфические для нежелательных клеток. В некоторых вариантах осуществления изобретение относится к применению не встречающихся в природе или сконструированных способами инженерии композиций, векторных систем или систем доставки, как описано в настоящем описании, для лечения, предотвращения или облегчения злокачественной опухоли. В некоторых вариантах осуществления изобретение относится к способу лечения, предотвращения или облегчения злокачественной опухоли, включая введение или индукцию не встречающихся в природе или сконструированных способами инженерии композиций, векторных систем или систем доставки, как описано в настоящем описании. Необходимо понимать, что система CRISPR-Cas предпочтительно нацелена на мишени, специфичные для злокачественных клеток. В некоторых вариантах осуществления изобретение относится к применению не встречающихся в природе или сконструированных способами инженерии композиций, векторных систем или систем доставки, как описано в настоящем описании, для лечения, предотвращения или облегчения инфекции клеток патогенами. В некоторых вариантах осуществления изобретение относится к не встречающимся в природе или сконструированным способами инженерии композициям, векторными системам или системам доставки, как описано в настоящем описании, для применения в лечении, предотвращении или облегчении инфекции клеток патогенами. В некоторых вариантах осуществления изобретение относится к способу лечения, предотвращения или облегчения инфекции клеток патогенами, включающему введение или индукцию не встречающихся в природе или сконструированных способами инженерии композиций, векторных систем или систем доставки, как описано в настоящем описании. Необходимо понимать, что система CRISPR-Cas предпочтительно нацелена на мишени, специфические для клеток, зараженных патогеном (например, мишень патогенного происхождения). В некоторых вариантах осуществления изобретение относится к применению не встречающихся в природе или сконструированных способами инженерии композиций, векторных систем или систем доставки, как описано в настоящем описании, для лечения, предотвращения или облегчения течения аутоиммунных заболеваний. В некоторых вариантах осуществления изобретение относится к не встречающимся в природе или сконструированным способами инженерии композициям, векторным системам или системам доставки, как описано в настоящем описании, для применения в лечении, предотвращении или облегчении аутоиммунных заболеваний. В некоторых вариантах осуществления изобретение касается способа лечения, предотвращения или облегчения аутоиммунных заболеваний, включающего введение или индукцию не встречающихся в природе или сконструированных способами инженерии композиций, векторных систем или систем доставки, как описано в настоящем описании. Необходимо понимать, что система CRISPR-Cas предпочтительно нацелена мишени, специфические для клеток, ответственных за аутоиммунные заболевания (например, определенные иммунные клетки).
Использование эффекторного белка нацеливания на РНК для обнаружении РНК
[336] Далее предусматривается, что эффекторный белок нацеливания на РНК может использоваться в анализе с использованием нозерн-блоттинга, который включает использование электрофореза для разделения образцов РНК по размеру. Эффекторный белок нацеливания на РНК может использоваться для специфичного связывания и обнаружения последовательности РНК-мишени.
[337] Эффекторный белок нацеливания на РНК может быть слит с флуоресцентным белком (таким как GFP) и использован для отслеживания локализации РНК в живых клетках. В частности, эффекторный белок нацеливания на РНК может быть инактивирован, так что он больше не сможет расщеплять РНК. В конкретных вариантах осуществления изобретения предусматривается, что может использоваться разделенный эффекторный белок нацеливания на РНК, в таком случае сигнал зависит от связывания обеих субъединиц для обеспечения более точной визуализации. Альтернативно, может использоваться разделенный флуоресцентный белок, который собирается, когда множественные комплексы эффекторного белка нацеливания на РНК связываются с транскриптом-мишенью. Далее предусматривается, что транскрипт нацеливается на множественные участки связывания вдоль мРНК, таким образом, флуоресцентный сигнал может усилить истинный сигнал и обеспечивать его фокальную идентификацию. В качестве другой альтернативы, флуоресцентный белок может быть собран из разделенного интеина.
[338] Эффекторные белки нацеливания на РНК, например, в подходящем случае используются для определения локализации РНК или определенных вариантов сплайсинга, количества транскрипта мРНК, повышения или понижения транскрипции и специфической диагностики заболеваний. Эффекторные белки нацеливания на РНК могут использоваться для визуализации РНК в (живых) клетках с использованием, например, флуоресцентной микроскопии или проточной цитометрии, такой как флуоресцентная сортировка клеток (FACS), которая обеспечивает высокую пропускную способность скрининга клеток и выделение живых клеток после сортировки. Далее, уровни экспрессии различных транскриптов могут быть оценены одновременно в условиях стресса, как например, ингибирование развития злокачественной опухоли с помощью воздействия на клетки молекулярных ингибиторов или гипоксических условий. Другой способ применения состоит в том, чтобы отследить транспорт транскриптов к синапсам во время нервного импульса с использованием двухфотонной лазерной микроскопии.
[339] В некоторых вариантах осуществления изобретения компоненты или комплексы согласно изобретению, как описано в настоящем описании, могут использоваться в мультиплексированной устойчивой к ошибкам флуоресцентной гибридизации in situ (MERFISH; Chen et al. Science, 2015, 348(6233)), например, такой как с (флуоресцентно) меченными эффекторами C2c2.
Мечение посредством apex in vitro
[340] Клеточные процессы зависят от сети молекулярных взаимодействий между белком, РНК и ДНК. Точное обнаружение взаимодействий белок-ДНК и белок-РНК является ключевым для понимания этих процессов. В технологии бесконтактной маркировки in vitro используется аффинная метка, соединенная, например, с фотоактивируемым зондом для мечения полипептидов и РНК вблизи белка-мишени или РНК-мишени in vitro. После облучения ультрафиолетом фотоактивируемая группа реагирует с белками и другими молекулами, находящимися в непосредственной близости от меченой молекулы, таким образом маркируя их. Меченые взаимодействующие молекулы могут впоследствии быть выделены и идентифицированы. Эффекторный белок нацеливания на РНК изобретения может, например, использоваться для нацеливания зонда на выбранную последовательность РНК.
[341] Эти способы использования также могут быть применены к модельным животным для визуализации in vivo соответствующих заболеваний или сложных для культивирования типов клеток.
Использование эффекторного белка нацеливании на РНК в РНК-оригами/в сборочных линиях in vitro - комбинаторике
[341] Под "РНК-оригами" понимаются наноразмерные сложенные структуры для создания двумерных или трехмерных конструктов с использованием РНК как интегрированного шаблона. Сложенная структура закодирована в РНК, таким образом, форма получающейся РНК определена синтезируемой последовательностью РНК (Geary, et al. 2014. Science, 345 (6198). pp. 799- 804). РНК-оригами может действовать как каркас для организации других компонентов, таких как белки, в комплексы. Эффекторный белок нацеливания на РНК изобретения может использоваться, например, для нацеливания на белки-мишени на РНК-оригами с использованием подходящей направляющей РНК.
[342] Эти способы использования также могут быть применены к модельным животным для визуализации in vivo соответствующих заболеваний или сложных для культивирования типов клеток.
Использование эффекторного белка нацеливания на РНК для выделения, очистки, обогащения или обеднения РНК определенными последовательностями
[343] Далее предусматривается, что эффекторный белок нацеливания на РНК в комплексе с РНК может использоваться для выделения и/или очистки РНК. Эффекторный белок нацеливания на РНК, может быть, например, присоединен к аффинной метке, которая может использоваться для выделения и/или очистки эффекторного белка нацеливания на РНК. Такие способы применения могут быть полезны, например, при анализе профилей экспрессии генов в клетках.
В конкретных вариантах осуществления изобретения может предусматриваться, что эффекторные белки нацеливания на РНК могут использоваться для нацеливания на конкретную некодирующую РНК (нкРНК), таким образом блокируя ее активность, что может быть применимо для анализа ее функции. В некоторых вариантах осуществления изобретения эффекторный белок, как описано в настоящем описании, может использоваться для специфического обогащения конкретной РНК (включая, но не ограничиваясь, увеличение стабильности и т.д.), или, наоборот, специфического обеднения конкретной РНК (такой как, без ограничения, например, конкретные варианты сплайсинга, изоформы и т.д.).
Изучение функции linc-РНК и других ядерных РНК
[344] Современные стратегии нокдауна РНК, например, siPHK, имеют недостаток, заключающийся главным образом в том, что они ограничиваются нацеливанием на цитозольные транскрипты, так как белок функционирует в цитозоле. Преимущество эффекторного белка нацеливания на РНК данного изобретения как внешней системы, которая не важна для функций клетки, состоит в том, что он может использоваться в любом компартменте клетки. Путем присоединения последовательности NLS к эффекторному белку нацеливания на РНК, он может быть направлен в ядро для нацеливания на ядерную РНК. Например, этот способ предусматривает исследование функции linc-РНК. Длинные межгенные некодирующие РНК (linсРНК) представляют собой слабоизученную область науки. У большинства linc-РНК есть не известные к настоящему времени функции, которые могут быть исследованы с использованием эффекторного белка нацеливания на РНК по изобретению.
Идентификация РНК-связывающих белков
[345] Идентификация белков, связанных с конкретной РНК, может быть полезна для понимания роли многих РНК. Например, многие linc-РНК ассоциируются с транскрипционными и эпигенетическими регуляторами для контроля транскрипции. Понимание, какие белки связываются с конкретной linc-РНК, может помочь выявить компоненты данного регуляторного процесса. Эффекторный белок нацеливания на РНК по изобретению может быть разработан для присоединения биотин-лигазы к определенному транскрипту, чтобы локально маркировать связанные белки биотином. Потом белки могут быть выделены и проанализированы при помощи масс-спектрометриии с целью идентификации.
Сборка комплексов на РНК и транспортировка субстрата
[346] Эффекторные белки нацеливания на РНК по изобретению могут далее использоваться для сборки комплексов на самой РНК. Это может быть достигнуто путем функционализации эффекторного белка нацеливания на РНК с помощью многих связанных белков (например, компонентов конкретного пути синтеза). Альтернативно, многие эффекторные белки нацеливания на РНК могут быть функционализированы путем связывания с различными белками и нацелены на ту же самую или смежную РНК-мишень. Полезное применение сборки комплексов на РНК состоит, например, в облегчении переноса субстрата между белками.
Синтетическая биология
[347] Создание биологических систем полезно, в том числе для разработки возможных применений в медицине. Предусматривается, что программируемые эффекторные белки нацеливания на РНК по изобретению могут быть использованы путем присоединения к разделенным белкам токсичных доменов для индукции программируемой клеточной гибели, например, с использованием связанной со злокачественной опухолью РНК в качестве транскрипта-мишени. Далее, в синтетических биологических системах на процессы, включающие белок-белковое взаимодействие, можно влиять путем, например, образования комплексов слияния с соответствующими эффекторами, такими как киназы или другими ферментами.
Сплайсинг белков: интеины
[348] Сплайсинг белка - посттрансляционный процесс, при котором полипептид, называемый интеином, катализирует свое собственное вырезание из окружающих его полипептидов, называемых экстеинами, с последующим лигированием экстеинов. Соединение двух или более эффекторных белков нацеливания на РНК, как описано в настоящем описании, на транскрипте-мишени может использоваться, чтобы направить высвобождение разделенного интеина (Topilina and Mills Mob DNA. 2014 Feb 4; 5(1):5), таким образом допуская прямое вычисление существования мРНК транскрипта и последующее высвобождение белкового продукта, такого как метаболический фермент или транскрипционный фактор (для последующего приведения в действие процессов транскрипции). Этот способ применения может быть особенно актуален в синтетической биологии (см. выше), или крупномасштабном биопроизводстве (производство продукта только при определенных условиях).
Индуцируемые, дозируемые и самоинактивирующиеся системы.
[349] В одном варианте осуществления слитые комплексы, включающие эффекторный белок нацеливания на РНК по изобретению и эффекторный компонент, разработаны, чтобы быть индуцируемыми, например, светоиндуцируемыми или химически индуцируемыми. Такая индуцируемость позволяет активировать эффекторный компонент в желаемый момент вовремя.
[350] Например, индуцируемость светом может быть достигнута путем разработки слитого комплекса, в котором для слияния используется образование пары CRY2PHR/CIBN. Эта система особенно полезна для индукции светом белковых взаимодействий в живых клетках (Konermann S, et al. Nature. 2013; 500:472-476).
[351] Например, химическая индуцируемость может быть достигнута путем разработки комплекса слияния, в котором используется образование пары FKBP/FRB (белок, связывающий FK506/FKBP, связывающий рапамицин). При использовании этой системы рапамицин необходим для связывания белков (в Zetsche et al. Nat Biotechnol. 2015; 33(2): 139-42 описано использование этой системы для Cas9).
[352] После введения в клетку в виде ДНК, эффекторный белок нацеливания на РНК по изобретению может быть далее изменен индуцируемыми промоторами для участия в тетрациклин- или доксициклин-контролируемой активации транскрипции (системы экспрессии Tet-On и Tet-Off), системах экспрессии генов, индуцируемых гормонами, таких как индуцируемая экдизоном и индуцируемая арабинозой система экспрессии генов. При введении в клетку в виде РНК, экспрессия эффекторного белка нацеливания на РНК может быть изменена с помощью рибопереключателя, который может выявлять низкомолекулярные соединения, такие как тетрациклин (как описано в Goldfless et al. Nucleic Acids Res. 2012; 40(9):e64).
[353] В одном варианте осуществления доставка эффекторного белка нацеливания на РНК по изобретению может быть модулирована для изменения количества белка или cr-РНК в клетке, таким образом, изменяя величину желаемого эффекта или любых нежелательных неспецифических эффектов.
[354] В одном из вариантов осуществления изобретения может быть разработан самоинактивирующийся эффекторный белкок нацеливания на РНК. При введении в клетку в виде РНК, как мРНК, так и терапевтической РНК (Wrobleska et al. Nat Biotechnol. 2015 Aug; 33(8): 839-841), они могут самостоятельно инактивировать экспрессию и последующие эффекты, разрушив собственную РНК, таким образом, может быть достигнуто сокращение времени нахождения и потенциальных нежелательных эффектов.
[355] Для других способов применения in vivo эффекторных белков нацеливания на РНК, как описано в настоящем описании, см. Mackay JP et al. (Nat Struct Mol Biol. 2011 Mar; 18(3):256-61), Nelles et al. (Bioessays. 2015 Jul; 37(7):732-9) и Abil Z and Zhao H (Mol Biosyst. 2015 Oct; 11(10):2658-65), которые включены в настоящее описание в качестве ссылки. В частности, следующие способы применения предусматриваются в некоторых вариантах осуществления изобретения, предпочтительно в некоторых вариантах осуществления при помощи каталитически неактивного C2c2: усиление трансляции (например, слияние C2c2 c факторами трансляции (например, слитые конструкции с eIF4)); подавление трансляции (например, направляющая РНК, нацеливающая на участки связывания рибосом); пропуск экзона (например, направляющие РНК, нацеливающие на участки доноров и/или акцепторов сплайсинга); включение экзона (например, направляющая РНК, нацеливающая на участки донора и/или акцептора сплайсинга конкретного экзона для их включения или C2c2, слитый с компонентами сплайсосомы (например, мяРНК U1); доступ к локализации РНК (например, соединение C2c2 с маркером (например, с EGFP)); изменение локализации РНК (например, слияние C2c2 с сигналом локализации (например, слияние с NLS или NES)); деградация РНК (в этом случае не предполагается использование каталитически неактивного C2c2, если предполагается активность C2c2, альтернативно, для увеличения специфичности, может использоваться разделенный C2c2); ингибирование функций некодирующей РНК (например, микроРНК), таких как деградация или связывание направляющей РНК с функциональными участками (возможно титрование в конкретных участках путем релокализации посредством слитых конструкций C2c2-сигнальная последовательность).
[356] Как описано в настоящем описании ранее и продемонстрировано в Примерах, функция C2c2 нечувствительна к 5'- или 3'-удлинению cr-РНК и к удлинению шпилечной структуры cr-РНК. Следовательно, это предусматривает, что шпилечные структуры MS2 и других доменов связывания могут быть добавлены к cr-РНК, не влияя на формирование комплекса и связывание с транскриптами-мишенями. Такие модификации cr-РНК для связывания различных эффекторных доменов применимы в использовании эффекторных белков нацеливания на РНК, описанных выше.
[357] Как продемонстрировано в Примерах, C2c2, в особенности LshC2c2, способен к опосредованию устойчивости к РНК-фагам. Следовательно, предусматривается, что C2c2 может использоваться для иммунизации, например, животных, людей и растений, против патогенов, содержащих только РНК, включая, но не ограничиваясь ими, вирус Эбола и вирус Зика.
[358] Авторы настоящего изобретения показали, что C2c2 может процессировать (расщеплять) его собственную последовательность. Это относится как к дикому типу белка C2c2, так и к мутантному белку C2c2, содержащему один или более мутантных аминокислотных остатков R597, H602, R1278 и H1283, таких как одна или более модификаций, отобранных из R597A, H602A, R1278A и H1283A. Следовательно, предусматривается, что множественные cr-РНК, разработанные для различных транскриптов-мишеней и/или способов применения, могут быть доставлены как единственная пре-cr-РНК или как единственный транскрипт, управляемый одним промотором. Такой способ доставки имеет то преимущество, что он существенно более компактный, обеспечивает более простые синтез и доставку в вирусных системах. Предпочтительно, нумерация аминокислот, описанная в настоящем описании, относится к белку LshC2c2. Будет понятно, что точные положения аминокислот могут отличаться для ортологов LshC2c2, что может быть адекватно определено с помощью выравнивания белка, как известно в данной области, и, как описано в настоящем описании в других пунктах.
[359] Особенности изобретения также охватывают способы и применения композиций и систем, описанных в настоящем описании, в генной инженерии, например, для изменения или манипулирования экспрессией одного или более генов или одного или более генных продуктов, в прокариотических или эукариотических клетках, in vitro, in vivo или ex vivo.
[360] В одном из аспектов изобретение относится к способам и композициям для направленного изменения, например, сокращения, экспрессии РНК-мишени в клетках. В рассматриваемых способах предусматривается система C2c2 по изобретению, которая вмешивается в транскрипцию, стабильность и/или трансляцию РНК.
[361] В некоторых вариантах осуществления изобретения эффективное количество системы C2c2 используется для расщепления РНК или иного ингибирования экспрессии РНК. В этом отношении система имеет применение, подобное миРНК и кшРНК, таким образом, она также может заменять такие способы. Способ включает, но не ограничивается этим, использование системы C2c2 вместо, например, интерферирующей рибонуклеиновой кислоты (такой как миРНК или кшРНК) или их матрицы транскрипции, например, ДНК, кодирующей кшРНК. Систему C2c2 вводят в клетку-мишень, например, путем введения млекопитающему, в котором находится клетка-мишень.
[362] Одним из преимуществ системы C2c2 по изобретению является ее специфичность. Например, тогда как полинуклеотидные системы интерферирующих рибонуклеиновых кислот (такие как миРНК или кшРНК) имеют проблемы в области разработки, стабильности и нецелевого связывания, система C2c2 изобретения может быть разработана с высокой специфичностью.
Дестабилизированный C2c2
[363] В некоторых вариантах осуществления изобретения эффекторный белок (фермент CRISPR; C2c2) согласно изобретению, как описано в настоящем описании, связан с или присоединен к домену дестабилизации (DD). В некоторых вариантах осуществления изобретения доменом дестабилизации (DD) является ER50. В некоторых вариантах осуществления изобретения соответствующим лигандом стабилизации для этого домена дестабилизации (DD) является 4HT. По существу, в некоторых вариантах осуществления изобретения одним по меньшей мере из одного домена дестабилизации (DD) является ER50, и его стабилизирующим лигандом - 4HT или CMP8. В некоторых вариантах осуществления изобретения доменом дестабилизации (DD) является DHFR50. В некоторых вариантах осуществления изобретения соответствующим лигандом стабилизации для этого домена дестабилизации (DD) является TMP. По существу, в некоторых вариантах осуществления изобретения одним по меньшей мере из одного домена дестабилизации (DD) является DHFR50, и его стабилизирующим лигандом - TMP. В некоторых вариантах осуществления изобретения доменом дестабилизации (DD) является ER50. В некоторых вариантах осуществления изобретения соответствующим лигандом стабилизации для этого домена дестабилизации (DD) является CMP8. Следовательно, CMP8 может быть альтернативным лигандом стабилизации для 4HT в системе ER50. В то время как возможно, что CMP8 и 4HT могут/должны использоваться в качестве конкурентов, некоторые типы клеток могут быть более восприимчивы к одному или другому из этих двух лигандов, и на основе этой заявки и знаний в данной области, квалифицированный специалист может использовать CMP8 и/или 4HT.
[364] В некоторых вариантах осуществления изобретения один или два домена дестабилизации (DD) могут быть присоединены к N-концу фермента CRISPR, и один или два домена дестабилизации (DD) могут быть присоединены к С-концу фермента CRISPR. В некоторых вариантах осуществления изобретения по меньшей мере два домена дестабилизации (DD) связаны с ферментом CRISPR, и домены дестабилизации (DD) одинаковы, т.е. гомологичны. Таким образом оба (или два или более) домена дестабилизации (DD) могут быть представлены ER50. Это предпочтительно в некоторых вариантах осуществления изобретения. Альтернативно, оба (или два или более) домена дестабилизации (DD) могут быть представлены DHFR50. Это также предпочтительно в некоторых вариантах осуществления изобретения. В некоторых вариантах осуществления изобретения по меньшей мере два домена дестабилизации (DD) связаны с ферментом CRISPR, и домены дестабилизации (DD) различны, т.е. гетерологичны. Таким образом. один из доменов дестабилизации (DD) может быть представлен ER50, в то время как один или более доменов дестабилизации (DD) или любого другого DD могут быть представлены DHFR50. Наличие двух или более доменов дестабилизации (DD), которые являются гетерологичными, может быть выгодным, поскольку оно обеспечивает больший уровень контроля деградации. Тандемное соединение более одного домена дестабилизации (DD) на N- или C-конце может увеличивать деградацию, и такое тандемное соединение может представлять собой, например, ER50-ER50-C2c2 или DHFR-DHFR-C2c2. Предусматривается, что высокий уровень деградации возможен в отсутствие любого из стабилизирующих лигандов, промежуточные уровни деградации возможны в случае отсутствия одного из стабилизирующих лигандов и присутствия другого стабилизирующего лиганда, в то время как низкие уровни деградации возможны в присутствии обоих (или двух или более) стабилизирующих лигандов. Контроль деградации С2с2 также возможен при наличии N-концевого домена дестабилизации (DD) ER50 и C-концевого домена дестабилизации (DD) DHFR50.
[365] В некоторых вариантах осуществления изобретения соединение фермента CRISPR с доменом дестабилизации (DD) включает линкер между DD и ферментом CRISPR. В некоторых вариантах осуществления изобретения линкер представляет собой GlySer. В некоторых вариантах осуществления изобретения фермент DD-CRISPR далее содержит по меньшей мере один сигнал ядерного экспорта (NES). В некоторых вариантах осуществления изобретения фермент DD-CRISPR включает два или больше сигналов ядерного экспорта (NES). В некоторых вариантах осуществления изобретения фермент DD-CRISPR включает по меньшей мере один сигнал ядерной локализации (NLS). Это может быть в дополнение к NES. В некоторых вариантах осуществления изобретения фермент CRISPR содержит, или по существу состоит из, или состоит из сигнала локализации (ядерного импорта или экспорта) или, в качестве составной части, линкера между ферментом CRISPR и доменом дестабилизации (DD). HA-метка или Flag-метка также могут входить в состав изобретения в качестве линкеров. Заявители используют NLS и/или NES в качестве линкеров и также используют глицин-сериновый линкер, такой как всего лишь (GGGGS)3.
[366] Дестабилизирующие домены полезны тем, что придают нестабильность широкому спектру белков, см., например, статью Miyazaki, J Am Chem Soc. Mar 7, 2012; 134(9): 3942-3945, включенную в настоящее описание в качестве ссылки. CMP8 или 4-гидрокситамоксифен может дестабилизировать домены. В общем смысле, показано, что чувствительный к температуре мутант DHFR млекопитающих (DHFRts), дестабилизирующий остаток по правилу N-конца, стабилен при пермиссивной температуре, но нестабилен при 37°C. Добавление метотрексата, высокоаффинного лиганда для DHFR млекопитающих, к клеткам, экспрессирующим DHFRts, частично ингибировало деградацию белка. Это демонстрирует, что небольшая молекула лиганда может стабилизировать белок, в ином случае предназначенный для деградации в клетках. Производное рапамицина использовалось, чтобы стабилизировать нестабильного мутанта по домену FRB mTOR (FRB*) и восстановить функцию слитой киназы, GSK-3β.6,7. Эта система продемонстрировала, что лиганд-зависимая стабильность представляет собой привлекательную стратегию регулирования функции специфичного белка в сложной биологической среде. Система контроля активности белка может включать DD, который становится функциональным, когда происходит комплементация убиквитином, вызванная индуцированной рапамицином димеризацией FK506-связывающего белка и FKBP12. Мутанты белка FKBP12 человека или белка ecDHFR могут быть разработаны таким образом, чтобы быть метаболически нестабильными в отсутствие их высокоаффинных лигандов, Shield-1 или триметоприма (TMP), соответственно. Эти мутанты являются возможными доменами дестабилизации (DD), полезными в практическом применении изобретения; нестабильность DD, слитого с ферментом CRISPR, приводит к деградации протеасомой белка CRISPR в составе всего соединенного белка. Shield-1 и TMP связываются с и стабилизируют DD дозозависимым способом. Лиганд-связывающий домен рецептора эстрогена (ERLBD, остатки 305-549 из ERS1) также может быть использован в качестве домена дестабилизации. Так как сигнальный путь рецептора эстрогена участвует в развитии ряда заболеваний, таких как рак молочной железы, сигнальный путь хорошо изучен, и получены многочисленные агонисты и антагонисты рецептора эстрогена. Таким образом, известны совместимые пары ERLBD и лекарственных средств. Есть лиганды, которые связывают мутантные, но не формы дикого типа ERLBD. При помощи одного из этих мутантных доменов, кодирующего три мутации (L384M, M421G, G521R)12, можно регулировать стабильность DD, полученного из ERLBD, с использованием лиганда, который не вмешивается в эндогенные эстроген-чувствительные сети. Дополнительная мутация (Y537S) может быть введена, чтобы далее дестабилизировать ERLBD и сделать его потенциальным кандидатом на роль DD. Этот тетра-мутант является предпочтительным для разработки DD. Мутантный ERLBD может быть присоединен к ферменту CRISPR в качестве DD для регулирования или нарушения его стабильности с помощью лиганда. Другой DD может быть создан с помощью метки размером 12 кДа (107 аминокислот) на основе мутантного белка FKBP, стабилизированного лигандом Shield-1; см., например, Nature Methods 5, (2008). Например, DD может быть модифицирован FK506-связывающим белком 12 (FKBP 12), который связывается с и обратимо стабилизируется синтетическим продуктом, биологически инертной небольшой молекулой, Shield-1; см., например, работы Banaszynski LA, Chen LC, Maynard-Smith LA, Ooi AG, Wandless TJ. A rapid, reversible, and tunable method to regulate protein function in living cells using synthetic small molecules. Cell. 2006; 126:995-1004; Banaszynski LA, Selimyer MA, Contag CH, Wandless TJ, Thome SH. Chemical control of protein stability and function in living mice. Nat Med. 2008; 14:1123-1127; Maynard-Smith LA, Chen LC, Banaszynski LA, Ooi AG, Wandless TJ. A directed approach for engineering conditional protein stability using biologically silent small molecules. The Journal of biological chemistry. 2007; 282:24866-24872; and Rodriguez, Chem Biol. Mar 23, 2012; 19(3): 391-398 - все из которых включены в настоящее описание в качестве ссылки и могут использоваться в практике изобретения для связывания выбранного DD с ферментом CRISPR. Как видно, в данной области известно множество DD, и DD может быть связан, например, слит, предпочтительно, с помощью линкера, с ферментом CRISPR, посредством чего DD может быть стабилизирован в присутствии лиганда и дестабилизирован в отсутствие лиганда, в результате чего фермент CRISPR полностью дестабилизируется, или DD может быть стабилизирован в отсутствие лиганда и дестабилизирован в присутствии лиганда; DD позволяет регулировать и контролировать фермент CRISPR, а следовательно, комплекс или систему CRISPR-Cas - "включать" или "выключать", если можно так выразиться, таким образом предоставляя средства для регулирования или контроля системы, например, в in vivo или in vitro среде. Например, когда целевой белок экспрессируется в качестве слитой конструкции с меткой DD, он дестабилизирован и подвергается быстрой деградации в клетке, например, в протеасомах. Таким образом, отсутствие стабилизирующего лиганда приводит к деградации белка Cas, связанного с DD. Когда новый DD слит с белком-мишенью, его нестабильность передается белку-мишени, приводя к быстрой деградации всего слитого белка. Пиковая активность иногда выгодна для Cas для уменьшения неспецифических эффектов. Таким образом, предпочтительны кратковременные вспышки высокой активности. Данное изобретение в состоянии обеспечить такие пики. В некоторых смыслах система индуцируема. В некоторых других смыслах, система подавляется отсутствием стабилизирующего лиганда и инициируется присутствием стабилизирующего лиганда.
Применение системы CRISPR, нацеленной на РНК, к растениям и дрожжам
Определения:
[367] В целом термин "растение" касается любых разнообразных фотосинтетических, эукариотических, одноклеточных или многоклеточных организмов царства Растения (Plantae), для которых характерен рост путем клеточного деления, наличие хлоропластов, и клеточных стенок, состоящих из целлюлозы. Термин "растение" включает однодольные и двудольные растения. Более конкретно, предполагается, что растения включают, но не ограничиваются ими, покрытосеменные и голосеменные растения, такие как акация, люцерна, амарант, яблоко, абрикос, артишок, ясень, спаржа, авокадо, банан, ячмень, бобы, свекла, береза, бук, ежевика, черника, брокколи, брюссельская капуста, капуста, канола, мускусная дыня, морковь, маниока, цветная капуста, кедр, хлебные злаки, сельдерей, каштан, вишня, китайская капуста, цитрусовые, клементин, клевер, кофе, кукуруза, хлопок, вигна (коровий горох), огурец, кипарис, баклажан, вяз, эндивий, эвкалипт, фенхель, инжир, ель, герань, виноград, грейпфрут, земляной орех, физалис, тсуга, гикори, кудрявая капуста (кале), киви, кольраби, лиственница, салат-латук, лук-порей, лимон, лайм, робиния, сосна, адиантум, маис, манго, клен, дыня, просо, гриб, горчица, орехи, дуб, овес, масличная пальма, бамия, лук, апельсин, декоративное растение или цветок или дерево, папайя, пальма, петрушка, пастернак, горох, персик, арахис, груша, сфагнум, перец, хурма, голубиный горох (каян), сосна, ананас, подорожник, слива, гранат, картофель, тыква, радиккио, редька, рапс, малина, рис, рожь, сорго, сафлор, ива, соя, шпинат, ель, патиссон, земляника, сахарная свекла, сахарный тростник, подсолнечник, батат, сладкая кукуруза, мандарин, чай, табак, помидор, деревья, тритикале, газонные травы, репа, виноградная лоза, грецкий орех, водяной кресс, арбуз, пшеница, ямс, тис и цукини. Термин растение также охватывает водоросли (Algae), которые являются, главным образом, фотоавтотрофами, объединенными, прежде всего, отсутствием корней, листьев и других органов, которые характеризуют высшие растения.
[368] Способы направленного изменения экспрессии генов, использующие систему нацеливания на РНК, как описано в настоящем описании, могут использоваться для придания желаемых свойств по существу любому растению. Большое разнообразие растений и систем растительных клеток с желаемыми физиологическими и агрономическими характеристиками, описанными в настоящем описании, может быть разработано с использованием конструкции из нуклеиновых кислот, описанных в настоящем описании, и различных упомянутых выше способов преобразования. В предпочтительных вариантах осуществления изобретения целевые растения и растительные клетки для инженерии включают, но не ограничиваются ими, однодольные и двудольные растения, такие как зерновые культуры, включая хлебные культуры (например, пшеница, кукуруза, рис, просо, ячмень), фруктовые культуры (например, помидор, яблоко, груша, клубника, апельсин), кормовые культуры (например, люцерна), овощные культуры (например, морковь, картофель, сахарная свекла, ямс), листовые овощные культуры (например, салат, шпинат); цветущие растения (например, петуния, роза, хризантема), хвойные деревья и (например, сосна, пихта, ель), растения, используемые в фиторемедиации (например, растения, накапливающие тяжелые металлы); масличные культуры (например, подсолнечник, рапс) и растения, используемые в экспериментальных целях (например, Arabidopsis). Таким образом, способы и системы CRISPR-Cas могут использоваться в широком спектре растений, например, в двудольных растениях, принадлежащих к следующим отрядам: Magnoliales, llliciales, Laurales, Piperales, Aristochiales, Nymphaeales, Ranunculales, Papaverales, Sarraceniaceae, Trochodendrales, Hamamelidales, Eucomiales, Leitneriales, Myricales, Fagales, Casuarinales, Caryophyllales, Batales, Polygonales, Plumbaginales, Dilleniales, Theales, Malvales, Urticales, Lecythidales, Violales, Salicales, Capparales, Ericales, Diapensales, Ebenales, Primulales, Rosales, Fabales, Podostemales, Haloragales, Myrtales, Cornales, Proteales, Santales, Rafflesiales, Celastrales, Euphorbiales, Rhamnales, Sapindales, Juglandales, Geraniales, Polygalales, Umbellales, Gentianales, Polemoniales, Lamiales, Plantaginales, Scrophulariales, Campanulales, Rubiales, Dipsacales и Asterales; способы и системы CRISPR-Cas могут использоваться в однодольных растениях, таких как растения, принадлежащие к следующим отрядам: Alismatales, Hydrocharitales, Najadales, Triuridales, Commelinales, Eriocaulales, Restionales, Poales, Juncales, Cyperales, Typhales, Bromeliales, Zingiberales, Arecales, Cyclanthales, Pandanales, Arales, Lilliales и Orchidales, или в растениях, принадлежащих к голосеменным, например, принадлежащих к отрядам: Pinales, Ginkgoales, Cycadales, Araucariales, Cupressales и Gnetales.
[369] Система CRISPR, нацеленная на РНК, и способы ее применения, описанные в настоящем описании, могут использоваться в широком диапазоне видов растений, включенных в неограниченный список двудольных, однодольных или голосеменных растений, принадлежащих к следующим родам: Atropa, Alseodaphne, Anacardium, Arachis, Beilschmiedia, Brassica, Carthamus, Cocculus, Croton, Cucumis, Citrus, Citrullus, Capsicum, Catharanthus, Cocos, Coffea, Cucurbita, Daucus, Duguetia, Eschscholzia, Ficus, Fragaria, Glaucium, Glycine, Gossypium, Helianthus, Hevea, Hyoscyamus, Lactuca, Landolphia, Linum, Litsea, Lycopersicon, Lupinus, Manihot, Majorana, Malus, Medicago, Nicotiana, Olea, Parthenium, Papaver, Persea, Phaseolus, Pistacia, Pisum, Pyrus, Prunus, Raphanus, Ricinus, Senecio, Sinomenium, Stephania, Sinapis, Solanum, Theobroma, Trifolium, Trigonella, Vicia, Vinca, Vitis, and Vigna; and the genera Allium, Andropogon, Eragrostis, Asparagus, Avena, Cynodon, Elaeis, Festuca, Festulolium, Heterocallis, Hordeum, Lemna, Lolium, Musa, Oryza, Panicum, Pannesetum, Phleum, Poa, Secale, Sorghum, Triticum, Zea, Abies, Cunninghamia, Ephedra, Picea, Pirns и Pseudotsuga.
[370] Системы CRISPR, нацеленные на РНК, и способы их применения могут также использоваться в широком спектре "водорослей" или "клеток водорослей"; включая, например, водоросли, отобранные из нескольких эукариотических филюмов, включая следующие: Rhodophyta (красные водоросли), Chlorophyta (зеленые водоросли), Phaeophyta (коричневые водоросли), Bacillariophyta (диатомовые водоросли), Eustigmatophyta и динофлагелляты, а также прокариотический филюм Cyanobacteria (сине-зеленые водоросли). Термин "водоросли" включает, например, водоросли, отобранные из следующих родов: Amphora, Anabaena, Ankistrodesmus, Botryococcus, Chaetoceros, Chlamydomonas, Chlorella, Chlorococcum, Cyclotella, Cylindrotheca, Dunaliella, Emiliana, Euglena, Hematococcus, Isochrysis, Monochrysis, Monoraphidium, Nannochloris, Nannochloropsis, Navicula, Nephrochloris, Nephroselmis, Nitzschia, Nodularia, Nostoc, Oochromonas, Oocystis, Oscillatoria, Pavlova, Phaeodactylum, Playtmonas, Pleurochrysis, Porphyra, Pseudoanabaena, Pyramimonas, Stichococcus, Synechococcus, Synechocystis, Tetraselmis, Thalassiosira и Trichodesmium.
[371] Часть растения, т.е. "растительная ткань" может быть обработана согласно способам по настоящему изобретению, чтобы получить растение c улучшенными свойствами. Понятие "растительная ткань" также включает растительные клетки. Термин "растительная клетка", как используют в настоящем описании, относится к отдельным единицам живого растения, как в интактном целом растении, так и в изолированной форме, выращенной в культуре тканей in vitro, на питательных средах или агаре, в суспензии на питательной среде или буфере или в качестве части более высокоорганизованных единиц, например, таких как растительная ткань, орган растения или целое растение.
[372] Термин "протопласт" относится к растительной клетке, защитная клеточная стенка которой была полностью или частично удалена, например, механическим или ферментативным способом, что привело к появлению интактной биохимически компетентной единице живого растения, которая способна заново сформировать клеточную стенку, пролиферировать, регенерировать и вырасти в целое растение при надлежащих условиях выращивания.
[373] Термин "трансформация" в широком смысле относится к процессу генетической модификации растения-хозяина с помощью введения ДНК посредством Agrobacteria или одного из ряда химических или физических способов. Как используют в настоящем описании, термин "растение-хозяин" относится к растениям, включая любые клетки, ткани, органы или потомство растений. Много подходящих растительных тканей или растительных клеток могут быть преобразованы и включают, но не ограничены, следующими: протопласты, соматические эмбрионы, пыльца, листья, саженцы, стебли, каллусы, столоны, микроклубни и побеги. Понятие "растительная ткань" также относится к любому клону такого растения, семени, потомству, черенкам, возникших в результате полового или вегетативного размножения, и потомкам любого из них, такие как черенки или семена.
[374] Термин "трансформированный", как используют в настоящем описании, относится к клетке, ткани, органу или организму, в которые была введена чужеродная конструкция, такая как молекула ДНК. Введенная молекула ДНК может быть интегрирована в ДНК клетки-реципиента, ткани, органа или организма, таким образом, что введенная молекула ДНК передается последующему потомству. В этих вариантах осуществления изобретения "трансформированная" или "трансгенная" клетка или растение также могут включать потомство клетки или растения и потомство, полученное с помощью искусственного разведения, использующего такое преобразованное растение в качестве родителя в скрещивании, и демонстрирующее измененный фенотип в результате присутствия введенной молекулы ДНК. Предпочтительно, трансгенное растение фертильно и способно к передаче введенной ДНК потомству посредством полового размножения.
[375] Термин "потомство", такое как потомство трансгенного растения, относится к тому, что порождается, происходит от или получено из растения или трансгенного растения. Введенная молекула ДНК также может быть временно введена в клетку реципиента, таким образом, что последующее потомство не наследует введенную молекулу ДНК и, таким образом, не считается "трансгенным". Соответственно, как используют в настоящем описании, "нетрансгенное" растение или растительная клетка является растением, не содержащим чужеродную ДНК, устойчиво интегрированную в его геном.
[376] Термин "промотор растения", как используют в настоящем описании, является промотором, способным к инициированию транскрипции в растительных клетках, независимо от того, происходит ли он из растительной клетки. Иллюстративные подходящие промоторы растений включают, но не ограничиваются ими, промоторы, полученные из растений, вирусов растения и бактерий, таких как Agrobacterium или Rhizobium, которые включают гены, экспрессируемые в растительных клетках.
[378] Как используют в настоящем описании, термин "дрожжевая клетка" относится к любой клетке гриба в родов Ascomycota и Basidiomycota. Дрожжевые клетки могут включать почкующиеся дрожжевые клетки, делящиеся дрожжевые клетки и клетки плесени. Не будучи ограниченным этими организмами, многие типы дрожжей, используемых в лабораторных и промышленных условиях, являются частью рода Ascomycota. В некоторых вариантах осуществления изобретения дрожжевая клетка является клеткой S. cerevisiae, Kluyveromyces marxianus или Issatchenkia orientalis cell. Другие дрожжевые клетки могут включать, но не ограничиваться ими, Candida spp. (например, Candida albicans), Yarrowia spp. (например, Yarrowia lipolytica), Pichia spp. (например, Pichia pastoris), Kluyveromyces spp. (например, Kluyveromyces lactis и Kluyveromyces marxianus), Neurospora spp. (например, Neurospora crassa), Fusarium spp. (например, Fusarium oxysporum), и Issatchenkia spp. (например, Issatchenkia orientalis, также именуемые Pichia kudriavzevii и Candida acidothermophilum). В некоторых вариантах осуществления изобретения клетка гриба является нитевидной клеткой гриба. Как используют в настоящем описании, термин "нитевидная клетка гриба" относится к любому типу клетки гриба, которая растет в виде нитей, т.е. гиф или мицелия. Примеры волокнистых клеток гриба могут включать, но не ограничиваться ими, Aspergillus spp. (например, Aspergillus niger), Trichoderma spp. (например, Trichoderma reesei), Rhizopus spp., (например, Rhizopus oryzae) и Mortierella spp. (например, Mortierella isabellina).
[379] В некоторых вариантах осуществления изобретения клетка гриба является промышленным штаммом. Как используют в настоящем описании, "промышленный штамм" относится к любому штамму клетки гриба, используемому в или выделенному из производственного процесса, например, производства продукта в коммерческих или промышленных масштабах. Понятие "промышленный штамм" может относиться к виду гриба, который, как правило, используется в производственном процессе, или оно может относиться к изоляту вида гриба, который также может использоваться в непромышленных целях (например, лабораторных исследованиях). Примеры производственных процессов могут включать брожение (например, в производстве продуктов питания или напитков), дистилляцию, производство биотоплива, производство различных соединений и полипептидов. Примеры промышленных штаммов могут включать, но не ограничиваться ими, JAY270 и АТСС4124.
[380] В некоторых вариантах осуществления изобретения клетка гриба является полиплоидной клеткой. Как используют в настоящем описании, понятие "полиплоидная" клетка может относиться к любой клетке, геном в которой присутствует больше, чем в одной копии. Понятие "полиплоидная клетка" может относиться к клетке, которая естественным образом существует в полиплоидном состоянии, или к клетке, полиплоидное состояние генома которой было вызвано искусственно (например, посредством специфического регулирования, изменения, инактивации, активации, или модификации мейоза, цитокинеза, или репликации ДНК). "Полиплоидная клетка" может относиться к клетке, весь геном которой является полиплоидным, или к клетке, которая является полиплоидом в конкретном геномном локусе-мишени. Чтобы не привязываться к теории, считается, что изобилие направляющих РНК может ограничивать скорость генной инженерии полиплоидных клеток в большей степени, чем гаплоидных клеток, и, таким образом, в способах использования системы CRISPR с белком С2с2, описанных в настоящем описании, могут использоваться преимущества определенного типа клетки гриба.
[381] В некоторых вариантах осуществления изобретения клетка гриба является диплоидной клеткой. Как используют в настоящем описании, понятие "диплоидная" клетка может относиться к любой клетке, геном которой присутствует в двух копиях. Диплоидная клетка может относиться к типу клетки, которая естественным образом существует в диплоидном состоянии, к клетке, диплоидное состояние генома которой было вызвано искусственно (например, посредством специфического регулирования, изменения, инактивации, активации или модификации мейоза, цитокинеза или репликации ДНК). Например, штамм S. cerevisiae, S228C, может находиться в гаплоидном или диплоидном состоянии. "Диплоидная" клетка может относиться к клетке, весь геном которой является диплоидным, или к клетке, которая является диплоидной в конкретном геномном локусе-мишени. В некоторых вариантах осуществления изобретения клетка гриба является гаплоидной клеткой. Как используют в настоящем описании, "гаплоидная" клетка может относиться к любой клетке, геном которой присутствует в одной копии. Гаплоидная клетка может относиться к типу клетки, которая естественно находится в гаплоидном состоянии, или к клетке, гаплоидное состояние генома которой было вызвано искусственно (например, посредством специфичного регулирования, изменения, инактивации, активации или модификации мейоза, цитокинеза или репликации ДНК). Например, штамм S. cerevisiae, S228C, может находиться в гаплоидном или диплоидном состоянии. "Гаплоидная" клетка может относиться к клетке, весь геном которой является гаплоидным, или к клетке, которая является гаплоидной в конкретном геномном локусе-мишени.
[382] Как используют в настоящем описании, "экспрессирующий вектор дрожжей" относится к нуклеиновой кислоте, которая содержит одну или более последовательностей, кодирующих РНК и/или полипептид, и может далее содержать любые желаемые элементы, которые управляют экспрессией нуклеиновой кислоты (кислот), а также любыми элементами, которые обеспечивают репликацию и сохранение вектора экспрессии в дрожжевой клетке. В данной области известно множество подходящих экспрессирующих векторов дрожжей и их особенностей; например, различные векторы и способы иллюстрированы в Yeast Protocols, 2nd edition, Xiao, W., ed. (Humana Press, New York, 2007) and Buckholz, R.G. and Gleeson, M.A. (1991) Biotechnology (NY) 9(11): 1067-72. Векторы дрожжей могут содержать, без ограничения, центромерную (CEN) последовательность, автономно реплицирующуюся последовательность (ARS), промотор, такой как промотор РНК-полимеразы III, функционально связанный с целевой последовательностью или геном, терминатор, такой как терминатор РНК-полимеразы III, ориджин репликации и маркерный ген (например, ауксотрофии, антибиотика или другие выбранные маркеры). Примеры экспрессирующих векторов для использования в дрожжах могут включать плазмиды, дрожжевые искусственные хромосомы, плазмиды 2μ, дрожжевые интегративные плазмиды, дрожжевые репликативные плазмиды, челночные векторы и эписомы.
Стабильная интеграция компонентов системы CRISPR, нацеленная на РНК, в геном растений и растительных клеток
[383] В конкретных вариантах осуществления изобретения предусматривается, что полинуклеотиды, кодирующие компоненты системы CRISPR, нацеленной на РНК, вводятся для стабильной интеграции в геном растительной клетки. В этих вариантах осуществления изобретения конструкция вектора трансформации или системы экспрессии может быть отрегулирована в зависимости от того, когда, где и при каких условиях направляющая РНК и/или ген(ы) нацеливания на РНК экспрессируются.
[384] В конкретных вариантах осуществления изобретения предусматривается введение компонентов системы CRISPR, нацеленной на РНК, стабильно в геномную ДНК растительной клетки. Дополнительно или альтернативно, предусматривается введение компонентов системы CRISPR, нацеленной РНК, для стабильной интеграции в ДНК органелл растений, таких как, но не ограниченных ими, пластиды, митохондрии или хлоропласты.
[385] Система экспрессии для стабильной интеграции в геном растительной клетки может содержать один или более следующих элементов: элемент промотора, который может использоваться для экспрессии фермента нацеливания на РНК и/или молекулы направляющей РНК в растительной клетке; 5'-нетранслируемые области для увеличения экспрессии; элемент интрона для дальнейшего увеличения экспрессии в определенных клетках, таких как клетки однодольного растения, сайт множественного клонирования, чтобы обеспечить удобные сайты рестрикции для вставки одной или более направляющих РНК и/или последовательностей генов нацеливания на РНК и других желаемых элементов; и 3'-нетранслируемые области для обеспечения эффективной терминации экспрессируемого транскрипта.
[386] Элементы системы экспрессии могут быть на одной или более конструкциях экспрессии, которые являются либо кольцевыми, такими как плазмида или вектор трансформации, либо линейными, такими как двойная спираль ДНК.
В конкретном варианте система экспрессии CRISPR, нацеленная на РНК, включает, по меньшей мере:
(a) нуклеотидную последовательность, кодирующую направляющую РНК (гРНК), которая гибридизируется с последовательностью-мишенью в растении, и где направляющая РНК включает направляющую последовательность и последовательность прямого повтора;
(b) нуклеотидную последовательность, кодирующую белок нацеливания на РНК,
где компоненты (a) или (b) расположены на одной и той же или на различных конструкциях, и посредством чего различные нуклеотидные последовательности могут находиться под контролем одного и того же или различных регуляторных элементов, действующих в растительной клетке.
[387] Конструкция (конструкции) ДНК, содержащая компоненты системы CRISPR, нацеленной на РНК, и, когда применимо, последовательность матрицы, может быть введена в геном растения, часть растения или растительную клетку рядом обычных способов. Процесс обычно включает этапы отбора подходящей клетки или ткани, введение конструкции (конструкций) в клетку или ткань и регенерацию растительных клеток или растений из клеток или тканей. В конкретных вариантах осуществления изобретения конструкция ДНК может быть введена в растительную клетку с использованием способов, таких как, но не ограничиваясь этим, электропорация, микроинъекция, аэрозольная инъекция протопластов растительной клетки, или конструкции ДНК могут быть введены непосредственно в растительную ткань с использованием биолистических способов, таких как бомбардировка частицами ДНК (см. также Fu et al., Transgenic Res, 2000 Feb; 9(1):11 -9). Бомбардировка клеток частицами основана на ускорении частиц, покрытых геном(генами)-мишенью, приводящем к проникновению частиц в протоплазму и типично стабильной интеграции в геном, (см., например, Klein et al., Nature (1987), Klein et al., Bio/Technology (1992), Casas et al., Proc. Natl. Acad. Sci. USA (1993)).
[388] В конкретных вариантах осуществления изобретения конструкции ДНК, содержащие компоненты системы CRISPR, нацеленной на РНК, могут быть введены в растение с помощью агробактериальной трансформации. Конструкции ДНК могут быть объединены с подходящими регионами фланкирования T-ДНК и введены в обычный вектор Agrobacterium tumefaciens. Чужеродная ДНК может быть включена в геном растений путем заражения растений или инкубирования протопластов растения с бактериями Agrobacterium, содержащими одну или более Ti-плазмид (индуцирующая опухоль плазмида) (см., например, Fraley et al., (1985), Rogers et al., (1987) и патент США № 5563055).
Промоторы растений
[389] Для обеспечения подходящей экспрессии в растительной клетке, компоненты системы C2c2 CRISPR, описанной в настоящем описании, как правило, помещены под контроль промотора растения, т.е. промотора, действующего в растительных клетках. Предусматривается использование различных типов промоторов.
[390] Конститутивный промотор растения - промотор, который способен экспрессировать открытую рамку считывания (ORF), которую он контролирует во всех или почти всех тканях растения во время всех или почти всех стадий развития растения (называется "конститутивной экспрессией"). Одним не ограничивающим примером конститутивного промотора является промотор 35S вируса мозаики цветной капусты. Настоящее изобретение предусматривает способы модификации последовательностей РНК, и как таковое также предусматривает регулирование экспрессии биомолекул растения. В конкретных вариантах осуществления данного изобретения таким образом предпочтительно поместить один или более элементов системы CRISPR, нацеленной на РНК, под контроль промотора, который может быть отрегулирован. Понятие "регулируемый промотор" относится к промоторам, которые управляют экспрессией генов не конститутивно, а регулируемым во времени и/или пространстве образом, и включает тканеспецифичные, тканепредпочтительные и индуцируемые промоторы. Различные промоторы могут управлять экспрессией гена в различных тканях или типах клеток, или на различных стадиях развития, или в ответ на различные условия окружающей среды. В конкретных вариантах осуществления изобретения один или более компонентов системы CRISPR, нацеленной на РНК, экспрессируется под контролем конститутивного промотора, такого как промотор 35S вируса мозаики цветной капусты. Тканепредпочтительные промоторы могут быть использованы для нацеливания на увеличенную экспрессию в определенных типах клеток в определенных тканях растения, например клеток сосудов в листьях или корнях или в определенных клетках семени. Примеры конкретных промоторов для использования в системе CRISPR, нацеленной на РНК, можно найти в Kawamata et al., (1997) Plant Cell Physiol 38:792-803; Yamamoto et al., (1997) Plant J 12:255- 65; Hire et al., (1992) Plant Mol Biol 20:207-18,Kuster et al., (1995) Plant Mol Biol 29:759-72, и Capana et al., (1994) Plant Mol Biol 25:681-91. Индуцибельные промоторы, обеспечивающие пространственно-временной контроль редактирования или экспрессии генов, могут использовать энергию. Форма энергии может включать, но не ограничиваться этим, звуковую энергию, электромагнитную радиацию, химическую энергию и/или тепловую энергию. Примеры индуцируемых систем включают тетрациклин-индуцируемые промоторы (Tet-On или Tet-Off), двухгибридные системы активации транскрипции, использующие небольшие молекулы (FKBP, ABA, и т.д.), или системы, индуцируемые светом (фитохромы, домены LOV или криптохромы), такие как эффектор транскрипции, индуцируемый светом (LITE), которые управляют изменениями в транскрипционной активности последовательность-специфическим образом. Компоненты индуцируемой светом системы могут включать фермент CRISPR, нацеленный на РНК, светочувствительный гетеродимер цитохрома (например, из Arabidopsis thaliana), и транскрипционный домен активации/репрессии. Другие примеры индуцируемых ДНК-связывающих белков и способов их применения приведены в US 61/736465 и US 61/721283, которые включены в настоящее описание в качестве ссылки в полном объеме.
[391] В определенных вариантах осуществления изобретения временная или индуцируемая экспрессия может быть достигнута при помощи, например, регулируемых химическими стимулами промоторов, т.е. таких, для которых применение экзогенного химического агента вызывает экспрессию генов. Модуляция экспрессии генов может также быть достигнута при использовании репрессируемых химическими стимулами промоторов, т.е. таких, для которых применение химического агента подавляет экспрессию генов. Такие индуцируемые химическими стимулами промоторы включают, не ограничиваются ими, промотор кукурузы ln2-2, активируемый антидотами бензолсульфонамидных гербицидов (De Veylder et al., (1997) Plant Cell Physiol 38:568-77), промотор кукурузы GST (GST-lI-27, W093/01294), активированный гидрофобными электрофильными соединениями, используемыми в общих гербицидах, наносимых до всхода урожая, и промотор табака PR 1 (Ono et al., (2004) Biosci Biotechnol Biochem 68:803-7), активируемый салициловой кислотой. Промоторы, активность которых регулируется антибиотиками, такие как индуцируемые тетрациклином и репрессируемые тетрациклином промоторы (Gatz et al, (1991) Mol Gen Genet 227:229-37; патенты США № 5814618 и 5789156) также могут быть использованы в рамках насоящего изобретения.
Перемещение к определенным органеллам и/или экспрессия в определенных органеллах растения
[392] Система экспрессии может включать элементы для транслокации в и/или экспрессии в определенной органелле растения.
Нацеливание в хлоропласты
[393] В конкретных вариантах осуществления изобретения предусматривается использование нацеленной на РНК системы CRISPR для того, чтобы специфически изменить экспрессию и/или трансляцию генов хлоропласта или обеспечить экспрессию в хлоропласте. С этой целью используются способы трансформации хлоропласта или компартментализации компонентов системы CRISPR, нацеленной РНК, в хлоропласте. Например, введение генетических модификаций в геноме пластида может снизить проблемы биологической безопасности, такие как перенос генов через пыльцу.
[394] Способы трансформации хлоропласта известны в данной области и включают баллистическую трансформацию с бомбардировкой частицами, обработку ПЭГ и микроинъекцию. Кроме того, можно использовать способы, включающие перемещение кассет трансформации от ядерного генома к пластиду, согласно описанию в WO 2010061186.
[395] Альтернативно этому, предусматривается нацеливание одного или более компонентов CRISPR, нацеленных на РНК, в хлоропласт растения. Это достигается за счет встраивания в конструкцию экспрессии последовательности, кодирующей транзитный пептида хлопласта (CTP) или транзитный пептид пластида, функционально связанный с 5'-концевой областью последовательности, кодирующей нацеленный на РНК белок. CTP удаляется на этапе процессинга во время перемещения в хлоропласт. Квалифицированному специалисту известно о нацеливания экспрессируемых белков в хлоропласт (см., например, Protein Transport into Chloroplasts, 2010, Annual Review of Plant Biology, Vol. 61: 157-180). В таких вариантах осуществления изобретения также желательно нацеливание одной или более направляющих РНК в хлоропласт растения. Способы и конструкции, которые могут использоваться для транслокации направляющей РНК в хлоропласт благодаря последовательности локализации в хлоропласте, описаны, например, в US 20040142476 и включены в настоящее описание в качестве ссылки. Такие изменения конструкций могут быть введены в системы экспрессии по настоящему изобретению с целью эффективного перемещения направляющей(их) РНК, нацеливающей на РНК.
Введение полинуклеотидов, кодирующих систему нацеливания CRISPR-РНК в водорослевые клетки
[396] Трансгенные водоросли (или другие растения, такие как рапс) могут быть особенно полезными для производства растительных масел или биотоплива, такого как спирты (особенно метанол и этанол) или другие продукты. Они могут быть модифицированы способами инженерии таким образом, чтобы экспрессировать или сверхэкспрессировать большие объемы масла или спиртов для использования в отраслях промышленности, использующих нефть или биотопливо.
[397] В US 8945839 описан способ модификации способами инженерии вида микроводорослей (клетки Chlamydomonas reinhardtii) с использованием Cas9. Используя подобную технику, способы с использованием описанной в настоящем описании нацеленной на РНК системы CRISPR могут быть применены для видов рода Chlamydomonas и других водорослей. В определенных вариантах осуществления изобретения нацеленный на РНК белок и направляющая(ие) РНК введены в водоросли, в которых они экспрессируются, с использованием вектора экспрессии нацеленного на РНК белка под контролем конститутивного промотора, такого как Hsp70A-Rbc S2 или бета-2-тубулина. Направляющая РНК необязательно может быть доставлена с использованием вектора, содержащего промотор T7. Альтернативно этому, в клетки водоросли может быть досталвена РНК, нацеливающая на мРНК, и транскрибированная in vitro направляющая РНК. Квалифицированному специалисту доступны протоколы электропорации, в том числе стандартный рекомендуемый протокол из набора GeneArt Chlamydomonas Engineering kit.
Введение полинуклеотидов, кодирующих компоненты нацеливания на РНК, в дрожжевые клетки
[398] В определенных вариантах осуществления изобретения изобретение относится к применению нацеленной на РНК системы CRISPR для редактирования РНК в дрожжевых клетках. Способы трансформации дрожжевых клеток, которые могут быть использованы для введения полинуклеотидов, кодирующих компоненты нацеленной на РНК системы CRISPR, известны квалифицированному специалисту и рассмотрены в Kawai et al., 2010, Bioeng Bugs. 2010 Nov-Dec, 1(6): 395-403). Неограничивающие примеры включают трансформацию дрожжевых клеток обработкой ацетатом лития (с возможностью дальнейшей обработки ДНК-переносчиком и ПЭГ), баллистического способа или электропорации.
Временная экспрессия компонентов нацеленной на РНК системы CRISPR в растениях и растительной клетке
[399] В определенных вариантах осуществления изобретения предусматривается, что направляющая РНК и/или ген нацеленного на РНК белка экспрессируется в растительной клетке. В таких вариантах осуществления изобретения система CRISPR, нацеленная на РНК, способна обеспечить модификацию РНК-мишеней только тогда, когда и направляющая РНК, и нацеленный на РНК белок присутствуют в клетке, причем в дальнейшем возможен контроль экспрессии генов. Поскольку экспрессия нацеленного на РНК фермента является временной, растения, регенерирующие из таких растительных клеток, как правило, не содержат чужеродной ДНК. В конкретных вариантах осуществления изобретения нацеленный на РНК фермент стабильно экспрессируется растительной клеткой и направляющая последовательность экспрессируется временно.
[400] В особенно предпочтительных вариантах осуществления изобретения компоненты системы CRISPR, нацеленной на РНК, могут быть введены в растительные клетки с использованием вирусного вектора растений (Scholthof et al. 1996, Annu Rev Pbytopathol. 1996; 34:299-323). В следующих конкретных вариантах осуществления указанный вирусный вектор является вектором ДНК-вируса. Например, геминивируса (geminivirus) (например, вирус листовой курчавости капусты, вирус желтой карликовости фасоли, вирус карликовости пшеницы, вирус листовой курчавости томата, вирус полосатости кукурузы, вирус листовой курчавости табака или вирус золотой мозаики томата), или нановируса (nanovirus) (например, вирус желтого некроза бобовых). В других конкретных вариантах указанный вирусный вектор является вектором РНК-вируса, например, тобравируса (tobravirus) (например, вирус погремковости табака, вирус табачной мозаики), потексвируса (potexvirus) (например, X-вирус картофеля) или гордеивируса (hordeivirus) (например, вирус полосатой мозаики ячменя). Геномы репликации вирусов растений являются неинтегральными векторами, которые представляет интерес с учетом избегания производства ГМО-растений.
[401] В некоторых вариантах осуществления вектор, используемый для временной экспрессии нацеленных на РНК конструкций CRISPR, может быть, в частности, вектором pEAQ, сконструированным специально для опосредованной Agrobacterium временной экспрессии в протопласте (Sainsbury F. et al., Plant Biotechnol J. 2009 Sep:7(7):682-93). Точное нацеливание на локализацию в геноме было продемонстрировано с использованием вектора на основе модифицированного вируса листовой курчавости капусты (CaLCuV) для экспрессии направляющих РНК в стабильно траснгенных растениях, экспрессирующих фермент CRISPR (Scientific Reports 5, Article number: 14926 (2015), doi: 10.1038/srepl4926).
[402] В конкретных вариантах осуществления изобретения фрагменты двухцепочечной ДНК, кодирующие направляющую РНК и/или ген нацеленного на РНК белка, могут быть временно введены в клетку растения. В таких вариантах осуществления внесенные двухцепочечные фрагменты ДНК присутствуют в количествах, достаточных для модификации молекул(ы) РНК в клетке, но не остаются после окончания рассматриваемого периода времени или после одного или более клеточных делений. Способы прямого переноса ДНК известны квалифицированному специалисту (см., например, Davev et al. Plant Mol Biol 1989 Sep; 13(3):273-85.)
[403] В других вариантах осуществления изобретения полинуклеотид, кодирующий нацеленный на РНК белок, вводят в растительную клетку с его последующей трансляцией и процессированием в клетке, синтезирующей этот белок в количестве, достаточном для изменения молекул(ы) РНК в клетке (в присутствии по меньшей мере одной молекулы направляющей РНК), но который не остается после окончания рассматриваемого периода времени или после одного или более клеточных делений. Способы введения мРНК в протопласт растения известны квалифицированному специалисту ((см., например, Gallie, Plant Cell Reports (1993), 13; 119-122). Также предусматриваются комбинации различных описанных выше способов.
Доставка компонентов CRISPR, нацеленных на РНК, в растительную клетку
[404] В конкретных вариантах осуществления изобретения требуется доставить один или более компонентов системы CRISPR, нацеленных на РНК, напрямую в растительную клетку. Это представляет интерес, среди прочего, для получения нетрансгенных растений (см. ниже). В некоторых вариантах осуществления изобретения, один или более нацеленных на РНК компонентов получен(ы) вне растения или растительной клетки и доставлен(ы) в клетку. Например, в некоторых вариантах осуществления изобретения нацеленный на РНК белок получен in vitro до введения в растительную клетку. Нацеленный на РНК белок может быть получен различными известными квалифицированному в данной области специалисту способами, в том числе рекомбинантным получением. После экспрессии нацеленный на РНК белок выделяют, подвергают рефолдингу в случае необходимости, очищают и необязательно из него удаляют меток для очистки, такой как His-метка. Когда неочищенный, частично очищенный или более полно очищенный нацеленный РНК белок получают, его можно вводить в растительную клетку.
[405] В определенных вариантах осуществления изобретения нацеленный на РНК белок объединен с направляющей молекулой РНК, нацеливающей на РНК-мишень, для образования предварительно собранного рибонуклеопротеина.
[406] Отдельные компоненты или сам предварительно собранный рибонуклеопротеин могут быть введены в растительную клетку путем электропорации, бомбардировки частицами, покрытыми генным продуктом, ассоциированным с нацеливанием на РНК, химической трансфекции или некоторых другими средств транспорта через клеточную мембрану. Например, была продемонстрирована трансфекция протопласта растения с предварительно собранным рибонуклеопротеином CRISPR с целью подтвердить модификацию генома растения (как описано в Woo et at. Nature Biotechnology, 2015; DQI: 1.0.1038/nbt.3389). Эти способы могут быть изменены с целью достижения направленной модификации молекул РНК в растениях.
[407] В конкретных вариантах осуществления изобретения нацеленная на РНК система CRISPR может быть введена в растительные клетки с использованием наночастиц. Компоненты, такие как белок, нуклеиновая кислота или их сочетание, могут быть загружены или упакованы в наночастицы и применены к растениям (как описано, например, в WO 2008042156 и US 20130185823). В частности, варианты осуществления изобретения включают наночастицы, в которые загружена или в которых содержится молекула(ы) ДНК, кодирующая белок, нацеленный на РНК, молекулы ДНК, кодирующие направляющую РНК, и/или выделенная направляющая РНК, как описано в WO 2015089419.
[408] Следующим средством введения одного или более компонентов нацеленных на РНК систем CRISPR в растительную клетку является использование проникающих в клетку пептидов (CPP). В соответствии с этим отдельные варианты осуществления изобретения включают композиции, содержащие проникающие в клетку пептиды, связанные с нацеленным на РНК белком. В конкретных вариантах осуществления настоящего изобретения нацеленный на РНК белок и/или направляющая(ие) РНК сопряжены с одним или более CPP для их эффективной доставки в протопласт растения (Ramakrishna (2014, Genome Res. 2014 Jun; 24(6):1020-7, для Cas9 в клетках человека). В других вариантах осуществления ген нацеленного на РНК белка и/или направляющая(ие) РНК кодируются одной или более кольцевыми или некольцевыми молекулой(ами), сопряженными с одним или более CPP для доставки в протопласт растения. Протопласты растения далее регенерируют в растительные клетки, а далее в растения. CPP в целом представляют собой короткие пептиды длиной менее 35 аминокислот полученные либо из белка, либо из химерных последовательностей, способных транспортировать биомолекулы через клеточную мембрану вне зависимости от рецепторов. CPP могут представлять собой катионные пептиды, пептиды, имеющие гидрофобные последовательности, амфипатические пептиды, пептиды, богатые пролином, и антимикробные последовательности, а также химерные или двухкомпонентные пептиды (Pooga and Langel, 2005). CPP способны проникать через биологические мембраны и при этом запускать движение различных биомолекул через мембраны клеток в цитоплазму и улучшать их внутриклеточное перемещение, тем самым облегчая взаимодействие биомолекулы с мишенью. Примеры CPP в числе прочих включают: Tat, ядерный белок-активатор транскрипции, необходимый для репликации вируса ВИЧ типа I, пенетратин, сигнальная пептидная последовательность фактора роста фибробластов (ФРФ, FGF) Капоши, сигнальная пептидная последовательность интегрина β3; последовательность пептида полиаргинина Args, богатые аргинином молекулярные транспортеры, пептид "sweet arrow".
РНК-мишень, предназначенная для использования в растениях, водорослях или грибах
[409] РНК-мишень, т.е. представляющая интерес РНК представляет собой РНК, подлежащую нацеливанию посредством настоящего изобретения, с последующим привлечением к и связыванием с нацеленным на РНК белком на представляющем интерес целевом участке РНК-мишени. РНК-мишень может представлять собой любую подходящей форму РНК. Последняя в некоторых вариантах осуществления изобретения может являться мРНК. В других вариантах осуществления изобретения РНК-мишень может включать транспортную РНК (тРНК) или рибосомную РНК (рРНК). В других вариантах осуществления изобретения РНК-мишень может включать интерферирующую РНК (РНК-i), микроРНК (миРНК), микропереключатели (microswitches), микрозимы, сателлитные РНК и РНК-содержащие вирусы. РНК-мишень может быть расположена в цитоплазме растительной клетки, или в ядре клетки, или в органелле растительной клетки, такой как митохондрия, хлоропласт или пластида.
[410] В отдельных вариантах осуществления изобретения нацеленная на РНК система CRISPR используется для расщепления РНК или подавления экспрессии РНК иным образом.
Использование нацеленной на РНК системы CRISPR для модулирования экспрессии генов растения через модулирование РНК
[411] Нацеленный на РНК белок может также быть использован вместе с подходящей направляющей РНК для нацеливания на экспрессию генов через контроль процессинга РНК. Контроль процессинга РНК может включать реакции процессирования РНК, такие как сплайсинг РНК, включая альтернативный сплайсинг; вирусную репликацию (в частности, вирусов растений, включая вироиды в растениях и биосинтез тРНК). Нацеленный на РНК белок в сочетании с подходящей направляющей РНК может также быть использован для контроля активации РНК (РНК-а). РНК-а вызывает стимуляцию экспрессии генов, в связи с чем контроль экспрессии генов может быть достигнут путем нарушения или уменьшения РНК-а и как следствие меньшей стимуляции экспрессии генов.
[412] Нацеленный на РНК эффекторный белок, описанный в настоящем описании, может в дальнейшем быть использован для противовирусной активности в растениях, в частности, в отношении РНК-содержащих вирусов. Эффекторный белок может быть нацелен на вирусную РНК с использованием подходящей направляющей РНК, избирательной в отношении выбранной последовательности РНК вируса. В частности, эффекторный белок может быть активной нуклеазой, расщепляющей РНК, в частности, одноцепочечную РНК. Следовательно, возможно использование нацеленного на РНК эффекторного белка в качестве противовирусного средства. Неограничивающими примерами вирусов, нейтрализация которых возможна благодаря этому, являются вирус табачной мозаики (TMV), вирус пятнистого увядания томатов (TSWV), вирус мозаики огурца (CMV), вирус картофеля Y (PVY), вирус мозаики цветной капусты (CaMV) (вирус RT), вирус шарки сливы (PPV), вирус мозаики костра (BMV) и вирус картофеля X (PVX).
[413] Примеры модулирования экспрессии РНК в растениях, водорослях или грибах как альтернативы для целенаправленной генной модификации описаны в настоящем описании далее.
[414] Предметом отдельного интереса является регулируемый контроль экспрессии генов через регулируемое расщепление мРНК. Он может быть достигнут за счет помещения элементов нацеливания на РНК под контроль регулируемых промоторов, как это описано в настоящем описании.
Использование системы CRISPR, нацеленной на РНК, для восстановления функциональности молекул тРНК
[415] Pring et al. описали редактирование РНК в митохондриях и хлоропластах растений, которое включает изменение последовательностей мРНК, кодирующих белки, отличные от кодируемых ДНК. (Plant Mol. Biol. (1993) 21 (6): 1163-1170. doi:10.1007/BF00023611). В некоторых вариантах осуществления изобретения элементы нацеленной на РНК системы CRISPR, специфически нацеленные на мРНК митохондрий и хлоропластов, могут быть введены в растение или клетку растения для экспрессии различных белков в таких органеллах растительных клеток, имитируя процессы, происходящие in vivo.
Использование нацеленной на РНК системы CRISPR как альтернативы РНК-интерференции для ингибирования экспрессии РНК
[416] Нацеленная на РНК система CRISPR имеет применения, сходные с ингибированием РНК или РНК-интерференцией, следовательно, может заменять эти способы. В некоторых вариантах осуществления такие способы по настоящему изобретению включают использование нацеленной на РНК системы CRISPR в качестве замены, например, участвующей в интерференции рибонуклеиновой кислоты (такой как малая интерферирующая миРНК, кшРНК или дцРНК). Примеры ингибирования экспрессии РНК в растениях, водорослях или грибах в качестве альтернативы для направленной генной модификации описаны далее.
Использование нацеленной на РНК системы CRISPR для контроля интерференции РНК
[417] Управление интерферирующей РНК или микроРНК может помочь уменьшить неспецифические эффекты (OTE), описанные в случае использования данных подходов, путем уменьшения продолжительности жизни интерферирующей РНК или микроРНК in vivo или in vitro. В отдельных вариантах осуществления изобретения РНК-мишень может включать интерферирующую РНК, т.е. РНК, вовлеченную в пути интерференции РНК, такую как кшРНК, миРНК и т.д. В других вариантах осуществления изобретения РНК-мишень может включать микроРНК или двухцепочечную РНК (дцРНК).
[418] В других отдельных вариантах осуществления в случае, если нацеленный на РНК белок и подходящая(ие) направляющая(ие) РНК селективно экспрессируются (например, селективно в пространстве или времени под контролем регулируемого промотора, например, специфического для ткани или фазы клеточного цикла промотора и/или энхансера), это может быть использовано для "защиты" клеток или систем (in vivo или in vitro) от РНК-i в таких клетках. Это может быть полезно для соседних тканей или клеток, в которых РНК-i не требуется или в целях сравнения с теми клетками или тканями, где эффекторный белок и подходящая направляющая молекула не экспрессируются (т.е. где РНК-i не контролируется и где она находится под контролем соответственно). Нацеленный на РНК белок может быть использован для контроля или связывания молекул, представляющих собой или включающих РНК, таких как рибозимы, рибосомы или рибопереключатели. В вариантах осуществления изобретения направляющая РНК может привлекать нацеленный на РНК белок к данным молекулам таким образом, чтобы нацеленный на РНК белок мог связываться с ними.
[419] Нацеленная на РНК система CRISPR, описанная в настоящем описании, может быть применена в области технологий РНК-i in planta без проведения избыточных экспериментов, согласно настоящему описанию, включая защиту от насекомых-вредителей, лечение заболеваний растений и изменение устойчивости к гербициду, а также в лабораторном анализе растения и для других применений (см., например, Kim et al., в Pesticide Biochemistry and Physiology (Impact Factor: 2.01). 01/2015; 120. DOI: 10.1016/j.pestbp.2015.01.002; Sharma et al. Academic Journals (2015), Vol. 12(18) pp 2303-2312); Green J.M, в Pest Management Science, Vol 70(9), pp 1351-1357), поскольку данное применение обеспечивает основу для рационального конструирования данной системы.
Использование нацеленной на РНК системы CRISPR для изменения рибопереключателей и контроля метаболической регуляции растений, водорослей и грибов
[420] Рибопереключатели (также известные как аптозимы) являются регуляторными участками матричной РНК, которые связывают низкомолекулярные соединения и в результате регулируют экспрессию генов. Данный механизм позволяет клетке чувствовать внутриклеточную концентрацию таких малых молекул. Конкретный рибопереключатель управляет близлежащим геном путем изменения транскрипции, трансляции или сплайсинга этого гена. Таким образом, в отдельных вариантах осуществления изобретения предусмотрен контроль активности рибопереключателя за счет использования нацеленного на РНК белка в сочетании с подходящей направляющей РНК для нацеливания на рибопереключатель. Это может происходить за счет расщепления или связывания с рибопереключателем. В отдельных вариантах осуществления предусмотрено уменьшение активности рибопереключателя. Недавно был охарактеризован рибопереключатель, связывающийся с тиаминпирофосфатазой (TPP) и описана его роль в регуляции биосинтеза тиамина в растениях и водорослях. Более того, оказалось, что этот элемент является ключевым регулятором первичного метаболизма растений (Bocobza and Aharoni, Plant J. 2014 Aug, 79(4):693-703. doi: 10.1111/tpj.12540. Epub 2014 Jun 17). Рибопереключатели TPP также найдены в определенных грибах, таких как Neurospora crassa, где они управляют альтернативным сплайсингом для получения в определенных условиях вышележащей открытой рамки считывания (uORF), таким образом, влияя на экспрессию расположенных в ниже генов (Cheah MT et al., (2007)Nature 447 (7143): 497-500. doi:10.1038/nature05769). Описанная в настоящем описании нацеленная на РНК система CRISPR может использоваться для управления эндогенной активностью рибопереключателя в растениях, водорослях или грибах и как таковая может изменять экспрессию расположенных ниже генов, которые она регулирует. В конкретных вариантах осуществления изобретения нацеленная на РНК система CRISPR может использоваться для анализа функции рибопереключателя in vivo или in vitro и для изучения его связи с метаболической сетью. В определенных вариантах осуществления изобретения нацеленная на РНК система CRISPR потенциально может использоваться для конструирования способами инженерии рибопереключателей в качестве метаболических сенсоров в растениях и платформах генного контроля.
Использование нацеленной на РНК системы CRISPR для скрининга РНК-интерференции в растениях, водорослях или грибах.
[421] В результате идентификации генных продуктов, нокдаун которых связан с изменениями в фенотипе, могут быть изучены биологические пути с выявлением составляющих с помощью скрининга с использованием РНК-интерференции. В конкретных вариантах осуществления изобретения контроль может проявляться во время самого скрининга с помощью белков Guide 29 или Guide 30 и подходящей направляющей РНК, как описано в настоящем описании, для прекращения или снижения активности РНК-интерференции при скрининге, тем самым восстанавливая активность (в которую ранее произведено вмешательство) генного продукта (путем прекращения или снижения интерференции/экспрессии).
Использование нацеленных на РНК белков для визуализации молекул РНК in vivo и in vitro.
[422] В некоторых вариантах осуществления изобретение относится к связывающей нуклеиновую кислоту системе. Гибридизацию РНК in situ с комплементарными зондами следует считать эффективным способом. Обычно для обнаружения нуклеиновых кислот путем гибридизации используются флуоресцентно меченые олигонуклеотиды ДНК. Увеличенная эффективность была достигнута благодаря использованию определенных модификаций, таких как "закрытые" нуклеиновые кислоты (LNA), однако остается необходимость в эффективных и регулируемых альтернативах. По сути, меченые элементы системы нацеливания РНК могут использоваться в качестве альтернативы эффективной и приспосабливаемой системы для гибридизации in situ.
Дополнительные применения нацеленной на РНК системы CRISPR в растениях и дрожжах
Использование нацеленной на РНК системы CRISPR при производстве биотоплива
[423] Под термином "биотопливо" в настоящем описании понимается альтернативное топливо, полученное из растений и других растительных ресурсов. Возобновляемое биотопливо может быть извлечено из органического вещества, энергия которого была запасена в ходе процесса фиксации углерода или получена посредством использования или конверсии биомассы. Эта биомасса может быть использована в качестве биотоплива или может быть переведена в форму удобных содержащих энергию веществ за счет термической, химической или биохимической переработки. Такая переработка позволит получить топливо в твердой, жидкой или газообразной форме. Существует два типа биотоплива: биоэтанол и биодизель. Биоэтанол получают в основном путем сбраживания углеводов - целлюлозы (крахмала), получаемых главным образом из кукурузы и сахарного тростника. Биодизель, с другой стороны, производится главным образом из масличных культур, таких как рапс, масличная пальма и соя. Биотопливо используется, главным образом, в качестве топлива для транспортных средств.
Улучшение свойств растения для производства биотоплива
[424] В конкретных вариантах осуществления способы с использованием нацеленной на РНК системы CRISPR, как описано в настоящем описании, используют для изменения свойств клеточной стенки с целью облегчения доступа ключевых агентов гидролиза и более эффективного высвобождения сахаров, используемых для брожения. В некоторых вариантах осуществления изобретения может быть модифицирован биосинтез целлюлозы и/или лигнина. Целлюлоза является главным компонентом клеточной стенки. Биосинтез целлюлозы и лигнина регулируются согласованно. Уменьшая долю лигнина в растении можно увеличить долю целлюлозы. В некоторых вариантах осуществления изобретения описанные в настоящем описании способы используются для снижения уровня биосинтеза лигнина в растении с целью увеличения доли сбраживаемых углеводов. Конкретнее, способы, описанные в настоящем описании, используются для снижения активности по меньшей мере первого гена биосинтеза лигнина, выбранного из группы, состоящей из 4-кумарат-3-гидроксилазы (C3H), фенилаланинаммонийлиазы (PAL), циннамат-4-гидроксилазы (C4H), гидрокслициннамоилттрансферазы (HCT), O-метилтрансферазы кофейной кислоты (COMT), коффеоил-КоА-3-O-метилтрансферазы (CCoAOMT), ферулат-5-гидроксилазы (F5H), дегидрогеназы циннамилового спирта (CAD), циннамоил-КоА-редуктазы (CCR), 4-кумароат-КоА-лигазы (4CL), монолигнол-лигнин-специфической гликозилтрансферазы и альдегиддегидрогеназы (ALDH), как описано в WO 2008064289 A2.
[425] В некоторых вариантах осуществления изобретения описанные в настоящем описании способы используются для производства биомассы растения, образующей более низкие уровни уксусной кислоты при сбраживании (см. также WO 2010096488).
Модификация дрожжей для производства биотоплива
[426] В конкретных вариантах осуществления изобретения предлагаемый нацеленный на РНК фермент используется для производства биоэтанола в рекомбинантных микроорганизмах. Например, нацеленные на РНК ферменты могут быть использованы для получения микроорганизмов, таких как дрожжи, для производства биотоплива или биополимеров из сбраживаемых сахаров и, возможно, для разложения лигноцеллюлозы, полученной из растений, извлеченных из отходов сельского хозяйства в качестве источника сбраживаемых сахаров. Конкретнее, изобретение относится к способам, в которых нацеленный на РНК комплекс CRISPR используется для изменения экспрессии эндогенных генов, необходимых для синтеза биотоплива, и/или изменения эндогенных генов, которые могут препятствовать синтезу биотоплива. Более конкретно, такие способы подразумевают стимуляцию экспрессии в микроорганизмах, таких как дрожжи, одной или более нуклеотидных последовательностей, кодирующих ферменты, участвующие в преобразовании пирувата в этанол или иной целевой продукт. В определенных вариантах осуществления такие способы обеспечивают стимуляцию экспрессии одного или более ферментов, позволяющих микроорганизмам разлагать целлюлозу, таких как целлюлаза. В других последующих вариантах осуществления нацеленный на РНК комплекс CRISPR, используется, чтобы подавить эндогенные метаболические процессы, конкурирующие с процессом производства биотоплива.
Модификация водорослей и растений для производства растительных масел или биотоплива
[427] Трансгенные водоросли или другие растения, такие как рапс, могут быть особенно востребованы при производстве растительных масел или биотоплива, например, такого как спирты (особенно метанол и этанол). Они могут быть целенаправленно разработаны для экспрессии или сверхэкспрессии больших объемов масел или спиртов для использования в отраслях промышленности, связанных с нефтью или биотопливом.
[428] В US 8945839 описан способ конструирования микроводорослей (клетки Chlamydomonas reinhardtii) при помощи Cas9. Используя подобные инструменты, способы с использованием описанной в настоящем описании нацеленной на РНК системы CRISPR могут быть применены к видам рода Chlamydomonas и другим водорослям. В отдельных вариантах осуществления изобретения нацеленный на РНК эффекторный белок и направляющая РНК молекула, введенные в водоросль, экспрессируются с помощью вектора, экспрессирующего нацеленный на РНК эффекторный белок под контролем конститутивного промотора, такой как S2 Hsp70A-Rbc или промотор бета-тубулина. Направляющая молекула РНК доставляется с использованием вектора, содержащего промотор T7. Альтернативно этому, транскрибированная in vitro направляющая РНК может быть доставлена в клетки водорослей. Методика электропорации соответствует стандартному рекомендуемому протоколу набора GeneArt Chlamydomonas Engineering kit.
Конкретные способы применения ферментов нацеливания на РНК в растениях
[429] В конкретных вариантах осуществления изобретения предполагается его использование в качестве терапии для удаления вирусов из растительных систем, поскольку оно позволяет расщеплять вирусную РНК. Предыдущие исследования, проведенные для систем человека, продемонстрировали успешное использование CRISPR для нацеливания на вирус гепатита С, содержащий одноцепочечную РНК (A. Price, et al., Proc. Natl. Acad. Sci, 2015). Эти способы могут также быть адаптированы для использования нацеленной на РНК системы CRISPR в растениях.
Улучшение свойств растений
[430] Настоящее изобретение также относится к растениям и дрожжевым клеткам, которые могут быть получены с помощью способов, описанных в настоящем описании. Растения с улучшенными свойствами, полученные описанными в настоящем описании способами, могут быть полезны для производства продуктов питания или кормов путем изменения экспрессии генов, обеспечивающих, в частности, устойчивость к вредителям, гербицидам, засухе, низким или высоким температурам, чрезмерному увлажнению и т.д.
[431] Растения с улучшенными свойствами, полученные описанными в настоящем описании способами, в особенности зерновые культуры и водоросли, могут быть произведены в качестве продуктов питания и кормов для обеспечения, например, более высокого содержания белков, углеводов, питательных веществ или витаминов по сравнения с таковыми для дикого типа. В этом отношении предпочтительны растения с улучшенными свойствами, в особенности бобовые и клубненосные.
[432] Улучшенные водоросли или другие растения, такие как рапс, могут быть особенно полезными для производства растительных масел или биотоплива, например такого, как спирты (особенно метанол и этанол). Они могут быть модифицированы способам инженерии для экспрессии или сверхэкспрессии высоких уровней масел или спиртов с целью использования в отраслях промышленности, связанных с нефтью или биотопливом.
[433] Также изобретение относится к улучшению составляющих частей растений. Такие части растения включают, но не ограничиваются ими, листья, стебли, корни, клубни, семена, эндосперм, яйцеклетки и пыльцу. Такие части растения, как предусматривается в рамках настоящего изобретения, могут быть жизнеспособными, нежизнеспособными, регенерируемыми и/или нерегенерируемыми.
[434] Также настоящее изобретение охватывает получение растительных клеток и растений, полученных согласно способам по изобретению. Гаметы, семена, зародыши, либо зиготические либо соматические, потомство или гибриды растений, несущих генетическую модификацию, которые получены способами традиционного скрещивания, также входят в рамки настоящего изобретения. Такие растения могут содержать гетерологическую или чужеродную последовательность ДНК, встроенную в последовательность-мишень или ее заменяющую. Альтернативно, такие растения могут содержать только одну модификацию (мутация, делеция, инсерция, замена) в одном или более нуклеотидах. По существу такие растения могут отличаться от исходных растений только наличием конкретной модификации.
[435] В одном из вариантов осуществления изобретения система C2c2 используется для разработки устойчивых к патогену растений, например за счет достижения устойчивости в отношении болезней, вызываемых бактериями, грибами или вирусами. В некоторых вариантах осуществления изобретения устойчивость к вредителям может быть достигнута для технических зерновых культур путем получения системы C2c2, съедаемой насекомым-вредителем, что приводит к его гибели. В одном из вариантов осуществления изобретения система C2c2 используется для придания устойчивости к факторам абиотической среды. В другом варианте осуществления изобретения система C2c2 используется для придания устойчивости к засухе, засолению, условиям низких или высоких температур. Younis et al. 2014, Int. J. Biol. Sci. 10; 1150 приводят обзор потенциальных мишеней для способов разведения растения, каждый из которых может быть скорректирован или улучшен за счет использования описанной здесь системы C2c2. Некоторые не ограничивающие примеры зерновых культур-мишеней включают Arabidopsis thaliana (L.) Heynh., Oryza sativa L., Prunus domestica L., Gossypium hirsutum L., Nicotiana rustica L., Zea mays L., Medicago sativa L., Nicotiana benthamiana Domin.
[436] В одном из вариантов осуществления изобретения система C2c2 используется для контроля над вредителями сельскохозяйственных культур. Например, активная в организме вредителя система C2c2 может быть экспрессирована в растении-хозяине или может быть доставлена непосредственно в мишень, например, с помощью вирусного вектора.
[437] В одном из вариантов осуществления изобретение относится к способу эффективного получения гомозиготных организмов из гетерозиготного исходного организма, отличного от человека. В одном из вариантов осуществления изобретения оно используется для селекции растений. В другом варианте осуществления изобретения оно используется в животноводстве. В таких вариантах осуществления изобретения гомозиготный организм, такой как растение или животное, получен путем предотвращения или подавления рекомбинации путем вмешательства в функционирование по меньшей мере одного гена-мишени, подверженного двухцепочным разрывам цепи, образованию пар хромосом и/или обмену цепи.
Применение белков C2C2 в оптимизированных функциональных системах нацеливания на РНК
[438] В одном аспекте изобретение относится к системе для специфической доставки функциональных компонентов в окружающую среду, содержащую РНК. Это может быть обеспечено с помощью системы CRISPR, включающей нацеленные на РНК эффекторные белки по настоящему изобретению, которые делают возможным точное нацеливание на РНК различных компонентов. В частности, такие компоненты могут быть активаторами или репрессорами, такими как активаторы или репрессоры трансляции РНК, деградации и т.д. Способы применения этой системы описаны в настоящем описании.
[439] Согласно одному своему варианту осуществление, изобретение относится к неприродной или сконструированной способами инженерии композиции, содержащей направляющую РНК, включающую направляющую последовательность, способную к гибридизации с последовательностью-мишенью в представляющем интерес геномном локусе в клетке, причем направляющая РНК модифицирована путем внесения инсерции в одну или более отдельную последовательность(и) РНК, которая связывает адаптерный белок. В определенных вариантах осуществления последовательности РНК могут связываться с одним или более адаптерным белком (например, аптамерами), причем каждый адаптерный белок ассоциирован с одним или более функциональными доменами. Для таких нацеленных на РНК ферментов C2c2, как описано в настоящем описании, продемонстрирована возможность модификации направляющей последовательности. В определенных вариантах направляющая РНК модифицирована путем внесения инсерции в отдельную(ые) последовательность(и) РНК 5'-концевого прямого повтора в самом прямом повторе или 3'-конца направляющей последовательности. В случае наличия более чем одного функционального домена они могут быть одинаковыми или различными, например, два одинаковых или два различных активатора или репрессора. В одном из вариантов изобретение относится к описанной в настоящем описании композиции, в которой один или более функциональных доменов соединены с нацеленным на РНК ферментом таким образом, что при связывании с РНК-мишенью функциональный домен имеет пространственную ориентацию, позволяющую функциональному домену работать согласно соответствующей ему функции. В одном аспекте изобретение относится к описанной в настоящем описании композиции, которая включает комплекс CRISPR-Cas, имеющий по меньшей мере три функциональных домена, по меньшей мере один из которых ассоциирован с нацеленным на РНК ферментом и по меньшей мере два из которых ассоциированы с гРНК.
[440] В соответствии с этим в одном аспекте настоящее изобретение относится к композиции неприродных или сконструированных способами инженерии комплексов CRISPR-Cas, содержащих направляющую РНК, как описано в настоящем описании, и фермент CRISPR, который является нацеленным на РНК ферментом, причем такой нацеленный на РНК фермент может необязательно иметь как минимум одну мутацию, так что нацеленный на РНК фермент имеет не более 5% нуклеазной активности фермента, не имеющего по меньшей мере этой одной мутации, и необязательно одну или более мутаций, затрагивающих одну или более последовательностей сигнала ядерной локализации. В определенных вариантах осуществления направляющая РНК дополнительно или альтернативно изменена таким образом, чтобы обеспечить сохранение связывания нацеленного на РНК фермента, но при этом предотвратить расщепление нацеленным на РНК ферментом (как подробно описано в настоящем описании).
[441] В конкретных вариантах осуществления нацеленный на РНК фермент является ферментом C2c2 с ослабленной нуклеазной активностью, составляющей по меньшей мере 97% или 100% таковой фермента С2с2, не имеющего по меньшей мере одной мутации. В одном из вариантов изобретение относится к описанной в настоящем описании композиции, в которой фермент C2c2 содержит две или более мутации. Такие мутации могут являться мутациями одного или более из следующих аминокислотных остатков: R597, H602, R1278 и H1283, к примеру, одна или более следующих мутаций: R597A, H602A, R1278A и H1283A для белка C2c2 Leptotrichia shahii или в соответствующих положениях его ортолога.
[442] В определенных вариантах осуществления изобретения описанная в настоящем описании система нацеливания на РНК, как описано в настоящем описании выше, содержит два или более функциональных доменов. В определенных вариантах осуществления изобретения такие два или более функциональных домена являются гетерологичными функциональными доменами. В определенных вариантах осуществления изобретения система включает адаптерный белок, который является слитым белком, включающим функциональный домен, причем слитый белок необязательно содержит линкер между адаптером белком и функциональным доменом. В определенных вариантах осуществления изобретения такой линкер включает линкер GlySer. Дополнительно или альтернативно этому, один или более функциональных доменов могут быть присоединены к эффекторному белку РНК посредством линкера, необязательно линкера GlySer. В конкретных вариантах осуществления изобретения один или более функциональных доменов присоединены к нацеленному на РНК ферменту через один или оба домена HEPN.
[443] В одном из вариантов изобретения описанная в настоящем описании композиция, в которой один или более функциональных доменов, связанных с адаптерным белком или нацеленным на РНК ферментом, является доменом, способным активировать или подавлять трансляцию РНК. В одном из вариантов осуществления изобретение относится к описанной в настоящем описании композиции, в которой по меньшей мере один из одного или более функциональных доменов связаны с адаптерным белком, и адаптерный белок имеет одну или более активностей, включающих метилазную активность, деметилазную активность, активность активации транскрипции, активность репрессии транскрипции, активность фактора терминации транскрипции, активность модификации гистона, активность расщепления РНК, активность интеграции ДНК, активность расщепления ДНК, или активность связывания нуклеиновой кислоты, или активность молекулярного выключателя или способность к индукции химическими стимулами или светом.
[444] В одной аспекте изобретение относится к описанной в настоящем описании композиции, включающей последовательность аптамера. В конкретных вариантах осуществления последовательность аптамера представляет собой две или более последовательности аптамера, специфичные к одному и тому же адаптерному белку. В одном аспекте изобретение относится к описанной в настоящем описании композиции, в которой последовательность аптамера представляет собой две или более последовательности аптамера, специфичные к различным адаптерным белкам. В одном из вариантов, изобретение относится к описанной в настоящем описании композиции, в которой адаптерный белок содержит MS2, PP7, Qβ, F2, GA, fr, JP501, M12, R17, BZ13, JP34, JP500, KU1, М11, MX1, TW18, VK, SP, FI, ID2, NL95, TW19, AP205, ϕCb5, ϕСb8г, ϕСb12г, ϕСb23г, 7s, PRR1. В соответствии с этим в определенных вариантах осуществления изобретения, аптамер выбран из связывающего белка, специфически связывающего любой один из упомянутых выше адаптерных белков. В одной из версий изобретение относится к описанной в настоящем описании композиции, в которой используемая клетка является эукариотической клеткой. В одном из вариантов изобретение относится к описанной в настоящем описании композиции, в которой такая эукариотическая клетка является клеткой млекопитающего, клеткой растения или дрожжевой клеткой, причем такая клетка млекопитающего может являться клеткой мыши. В одном из вариантов изобретение относится к описанной в настоящем описании композиции, в которой такая клетка млекопитающего является клеткой человека.
[445] В одном из аспектов изобретение относится к описанной в настоящем описании выше композиции, в которой содержится более одной гРНК и гРНК нацелены на различные последовательности, причем при использовании такой конструкции имеет место мультиплексирование. В одном из аспектов изобретение относится к композиции, в которой содержится более одной гРНК, измененной путем инсерции определенной последовательности(ей) РНК, которая связывает один или более адаптерных белков.
[446] В одном из аспектов изобретение относится к описанной в настоящем описании композиции, в которой содержится один или более адаптерных белков, связанных с одним или более функциональными доменами, и такие белки связаны с отдельной последовательностью(ями) РНК, вставленной(ыми) в направляющую(ие) РНК.
[447] В одном аспекте изобретение относится к описанной в настоящем описании композиции, в которой направляющая РНК изменена так, чтобы иметь по меньшей мере одну не кодирующую функциональную шпилечную структуру; например, в которой по меньшей мере одна не кодирующая функциональная шпилечная структура является репрессивной; например, в которой по меньшей мере одна не кодирующая функциональная шпилечная структура включает Alu-повтор.
[448] В одном аспекте изобретение относится к модификации экспрессии генов, включающей введение хозяину или экспрессию в хозяине in vivo одной или более конструкций, как описано в настоящем описании.
[449] В одном из аспектов изобретение относится к описанному в настоящем описании способу, включающему доставку композиции или молекулы (молекул) нуклеиновой кислоты, кодирующей таковую, причем указанная молекула(ы) нуклеиновой кислоты функционально связана с регуляторной(ыми) последовательностью(ями) и экспрессируется in vivo. В одном из вариантов изобретение относится к способу, описанному в настоящем описании, в котором экспрессия осуществляется in vivo в лентивирусе, аденовирусе или AAV.
[450] В одном из аспектов изобретение относится к линии клеток млекопитающего, как описано в настоящем описании, причем такая клеточная линия необязательно может быть линией клеток человека или линией клеток мыши. В одном из аспектов изобретение относится к трансгенной модели млекопитающего, необязательно мыши, причем такая модель трансформирована обсуждаемой в настоящем описании композиции или является потомством указанного трансформанта.
[451] В одном из аспектов изобретение относится к молекуле(ам) нуклеиновой кислоты, кодирующей направляющую РНК молекулу или нацеленный на комплекс CRISPR-Cas или композицию, описанную в настоящем описании. В одном из вариантов изобретение относится к вектору, включающему молекулу нуклеиновой кислоты, кодирующую направляющую РНК (гРНК), включающую последовательность направляющей молекулы, способной к гибридизации с последовательностью-мишенью в геномном локусе-мишени в клетке, где прямой повтор направляющей РНК изменен вставкой определенной последовательности(ей) РНК, которая связывает(ют) два или более адаптерных белка, причем каждый адаптерный белок связан с одним или более функциональными доменами; или направляющая РНК изменена таким образом, чтобы иметь по меньшей мере одну некодирующую функциональную шпилечную структуру. В одном из аспектов изобретение обеспечивает вектор(ы), включающий(ие) молекулу(ы) нуклеиновой кислоты, кодирующую(ие) неприродную или сконструированную способами инженерии композицию комплекса CRISPR-Cas, включающего направляющую РНК, обсуждаемую в настоящем описании, и фермент нацеливания на РНК, где необязательно фермент нацеливания на РНК включает по меньшей мере одну мутацию, так что фермент нацеливания РНК имеет не более 5% нуклеазной активности фермента нацеливания РНК, не имеющего такой по меньшей мере одной мутации, и необязательно одну или более последовательностей сигнала ядерной локализации. В одном из вариантов изобретения вектор, кроме того, может включать регуляторный элемент(ы), активный в эукариотической клетке, функционально связанный с молекулой нуклеиновой кислоты, кодирующей направляющую РНК (гРНК), и/или молекулой нуклеиновой кислоты, кодирующей фермент нацеливания на РНК, и/или дополнительной(ые) последовательностью(и) ядерной локализации.
[452] В одном аспекте изобретение относится к набору, содержащему один или более компонентов, описанных в настоящем описании выше. В некоторых вариантах осуществления изобретения такой набор включает векторную систему, описанную выше и инструкции по использованию набора.
[453] В одном аспекте изобретение относится к способу скрининга приобретения функции (GOF) или потери функции (LOF) или скрининга некодирующих РНК или потенциальных регуляторных областей (например, энхансеров, генов-репрессоров), включающему клеточную линию, как описано в настоящем описании, или клетки описанной в настоящем описании модели, содержащие или экспрессирующие фермент нацеливания на РНК, и введение композиции, как описано в настоящем описании, в клетки клеточной линии или модели, причем направляющая РНК включает активатор или репрессор, и мониторинг приобретения функции (GOF) или потери функции (LOF), соответственно, для тех клеток, в которых введенная направляющая РНК включает активатор, или для тех клеток, в которых введенная направляющая РНК включает репрессор.
[454] В одном аспекте изобретение относится к библиотеке неприродных или сконструированных способами инженерии композиций, каждая из которых содержит направляющую РНК (гРНК) системы CRISPR, нацеленной на РНК, включающую последовательность направляющей молекулы, способной к гибридизации с последовательностью РНК-мишенью в клетке-мишени, фермент нацеливания на РНК, где фермент нацеливания на РНК включает по меньшей мере одну мутацию, так что фермент нацеливания на РНК имеет не более 5% нуклеазной активности фермента нацеливания на РНК, не имеющего такой по меньшей мере одной мутации, и направляющая РНК изменена путем вставки определенной последовательности(ей) РНК, которая связывается с одним или более адаптерными белками, причем адаптерный белок ассоциирован с одним или более функциональными доменами, и где гРНК включают полногеномную библиотеку, содержащую множество направляющих РНК (гРНК), нацеливающих на РНК. В одном аспекте изобретение относится к описанной в настоящем описании библиотеке, где фермент, нацеленный на РНК, имеет сниженную нуклеазную активность, составляющую по меньшей мере 97% или 100% от таковой фермента, нацеленного РНК, не имеющего такой по меньшей мере одной мутации. В одном из аспектов изобретение относится к библиотеке, как описано в настоящем описании, в которой адаптерный белок является слитым белком, содержащим функциональный домен. В одном из вариантов изобретение относится к библиотеке, как описано в настоящем описании, в которой гРНК не изменена вставкой определенной последовательности(ей), которая(ые) связывает(ют) один или более адаптерных белков. В одном из аспектов изобретение относится к библиотеке, как описано в настоящем описании, в которой один, два или более функциональных домена связаны с ферментом нацеливания на РНК. В одном из аспектов изобретение относится к библиотеке, как описано в настоящем описании, в которой популяция клеток является популяцией эукариотических клеток. В одном из аспектов изобретение относится к библиотеке, как описано в настоящем описании, в которой такая эукариотическая клетка представляет собой клетку млекопитающего, растительную клетку или дрожжевую клетку. В одном из аспектов изобретение относится к библиотеке, как описано в настоящем описании, в которой такая клетка млекопитающих является клеткой человека. В одном из аспектов изобретение относится к библиотеке, как описано в настоящем описании, в которой такая популяция клеток является популяцией эмбриональных стволовых клеток (ES).
[455] В одном из аспектов изобретение относится к библиотеке, как описано в настоящем описании, с возможностью нацеливания приблизительно на 100 или более последовательностей РНК. В одном из аспектов изобретение относится к библиотеке, как обсуждается в настоящем описании, с нацеливанием приблизительно на 1000 или более последовательностей РНК. В одном из аспектов изобретение относится к библиотеке, как описано в настоящем описании, с нацеливанием приблизительно на 20000 или более последовательностей РНК. В одном из вариантов изобретение относится к библиотеке, как описано в настоящем описании, с нацеливанием, охватывающим весь транскриптом. В одном из вариантов изобретение относится к библиотеке, как описано в настоящем описании, с нацеливанием на набор последовательностей-мишеней, связанных с существенным или желательным процессом. В одном из вариантов осуществления изобретение относится к библиотеке, как описано в настоящем описании, в которой таким процессом является иммунный процесс. В одном из своих воплощений изобретение относится к библиотеке, как описано в настоящем описании, в которой такой процесс представляет собой процесс клеточного деления.
[456] В одной из версий изобретение относится к способу получения модельной эукариотической клетки, содержащей ген с измененной экспрессией. В некоторых вариантах осуществления изобретения под геном заболевания понимается любой ген, связанный с увеличением риска наличия или развития заболевания. В некоторых вариантах осуществления изобретения такой способ включает (a) введение одного или более векторов, кодирующих компоненты системы, описанной в настоящем описании, в эукариотическую клетку и (b) позволение комплексу CRISPR связывать РНК- или ДНК-мишень для изменения экспрессии гена, в результате получая модельную эукариотическую клетку с измененной экспрессией генов.
[457] Структурная информация, приведенная в настоящем описании, позволяет исследовать взаимодействия направляющей РНК и нацеленного на РНК фермента, делая возможным конструирование или изменение направляющей РНК для оптимизации функций всей системы CRISPR-Cas в целом. Например, направляющая РНК может быть удлинена без препятствования белку нацеливания на РНК за счет вставки адаптерных белков, которые могут связывать РНК. Эти адаптерные белки могут далее привлекать эффекторные белки или слитые конструкции, включающие один или более функциональных доменов.
[458] Аспектом изобретения является включение вышеупомянутых элементов в единую композицию или в отдельные композиции. Эти композиции преимущественно могут быть применены у хозяина для индукции функционального эффекта на геномном уровне.
[459] Квалифицированному специалисту будет понятно, что модификации направляющей РНК, которые допускают связывание адаптера+функционального домена, но не надлежащее взаиморасположение адаптера+функционального домена (например, из-за пространственных затруднений в трехмерной структуре комплекса CRISPR), являются нежелательными модификациями. Одна или более измененная(ые) направляющая(ие) молекула(ы) может быть изменена(ы) введением определенной(ых) последовательности(ей) на 5'-конце прямого повтора в самом прямом повторе или на 3'-конце направляющей последовательности.
[460] Измененная направляющая РНК, инактивированный фермент нацеливания на РНК (с функциональными доменами или без них) и связывающий белок с одним или более функциональным(и) доменом(ами), могут находиться в композиции по отдельности и доставляться в организм хозяина по отдельности или вместе. Альтернативно этому, такие компоненты могут быть доставлены в организм хозяина в единой композиции. Введение в организм хозяина может быть выполнено с использованием вирусных векторов, известных квалифицированному специалисту или описанных в настоящем описании для доставки в организм хозяина (например, лентивирусного вектора, аденовирусного вектора, вектора AAV). Как поясняется в настоящей заявке, использование различных селективных маркеров (например, для селекции лентивирусной гРНК) и концентраций направляющей РНК (например, зависящих от того, используются ли множественные гРНК) может быть преимущественным для обеспечения лучшего эффекта.
[461] Используя описанные композиции квалифицированный в данной области специалист может предпочтительно и специфично осуществить нацеливание на один или более локусов с одинаковыми или различными функциональными доменами для индукции одного или более геномных событий. Такие композиции могут быть использованы во множестве способов скрининга в библиотеках в клетках и функциональном моделировании in vivo (например, активация генов linc-РНК и идентификация функции; моделирование приобретения функции, моделирование потери функции; использование композиции по изобретению для получения клеточных линий и трансгенных животных в целях оптимизации и скрининга).
[462] Настоящее изобретение относится к использованию конструкций по настоящему изобретению для выявления и использования требующих определенных условий или индуцируемых событий нацеливания на РНК (см., например, Platt et al., Cell (2014), http://dx.doi.Org/T0.1016/j.cell.2014.09.014, или цитируемые в настоящем описании патентные публикации PCT, такие как WO 2014/093622 (PCT/US2013/074667), которые не являются уровнем техники для настоящего изобретения или заявки). Например, клетка-мишень включает нацеленный на РНК фермент CRISRP, кондициональный или индуцируемый (например, в форме Cre-зависимых конструкций), и/или адаптерный белок, кондициональный или индуцируемый, и при экспрессии вектора, введенного в клетку-мишень, происходит индукция или начало состояния экспрессии фермента нацеливания на РНК и/или адаптера в клетке-мишени. В случае применения концепции и композиции по изобретению с известным способом получения комплекса CRISPR, индуцируемая экспрессия генов, измененная функциональными доменами, также является аспектом настоящего изобретения. Альтернативно, адаптерный белок может быть предоставлен в качестве кондиционального или индуцибельного элемента с кондициональным или индуцибельным нацеленным на РНК белком для получения эффективной модели для скрининга, который преимущественно требует только минимального моделирования и внедрения специфических гРНК для широкого круга применений.
Направляющая РНК по изобретению, содержащая "мертвую" направляющую последовательность
[463] В одном возможном варианте осуществления изобретение относится к направляющим последовательностям, модифицированным таким образом, который позволяет образование комплекса CRISPR и успешное связывание с мишенью, в то же время не допуская продуктивной нуклеазной активности (т.е. без нуклеазной активности/без инсерционно/делеционной (indel) активности). Для пояснения такие измененные последовательности направляющей молекулы называются "мертвыми направляющими молекулами" или "мертвыми направляющими последовательностями". Такие мертвые направляющие молекулы или мертвые направляющие последовательности могут рассматриваться как каталитически неактивные или конформационно неактивные в отношении нуклеазной активности. Действительно, мертвые последовательности направляющей молекулы могут недостаточно активно участвовать в продуктивном попарном связывании оснований с учетом способности усиливать каталитическую активность или различать специфическую и неспецифическую активность связывания. В кратком изложении, анализ включает синтез РНК-мишени CRISPR и направляющих РНК, содержащих нарушения комплементарности с РНК-мишенью, комбинируя их с нацеленным на РНК ферментом и анализируя расщепление в гелях на основе присутствия полос, образованных продуктами расщепления, и проводя количественную оценку расщепления на основе относительной интенсивности полос.
[464] Следовательно, в связанном с этим аспекте изобретение относится к неприродной или сконструированной способами инженерии композиции нацеленной на РНК системы CRISPR-Cas, включающей функциональное нацеливание на РНК, как описано в настоящем описании, и направляющую РНК (гРНК), причем направляющая РНК включает мертвую последовательность направляющей молекулы, посредством чего гРНК способна к гибридизации с последовательностью-мишенью, таким образом, что нацеленная на РНК система CRISPR-Cas направляется к представляющему интерес геномному локусу-мишени в клетке без поддающейся обнаружению активности расщепления немутантного нацеленного на РНК фермента такой системы. Следует понимать, что любая из гРНК согласно изобретению, как описано в настоящем описании, может использоваться в качестве мертвых направляющих РНК/направляющих РНК, включающих мертвую направляющую последовательность, как описано в настоящем описании ниже. Любой из способов, продуктов, композиций и применений, как описано в настоящем описании, в равной степени применим к мертвым направляющим РНК/направляющим РНК, включающим мертвую направляющую последовательность, как подробно изложено ниже. Посредством дальнейшего руководства предусматриваются следующие конкретные аспекты и варианты осуществления изобретения.
[465] Способность мертвой направляющей последовательности обеспечивать специфичное к последовательности связывание комплекса CRISPR с последовательностью РНК-мишени может быть оценена любым подходящим способом анализа. Например, компоненты системы CRISPR, достаточные для формирования комплекса CRISPR, включая тестируемую мертвую направляющую последовательность, могут быть доставлены в клетку, имеющую соответствующую последовательность-мишень, например, путем трансфекции векторами, кодирующими компоненты последовательности CRISPR, с последующим установлением участков предпочтительного расщепления в последовательности-мишени. В частности, расщепление последовательности полинуклеотида РНК-мишени может быть оценено в пробирке, при использовании последовательности-мишени, компонентов комплекса CRISPR, включая тестируемую мертвую направляющую последовательность и контрольную направляющую последовательность, отличающуюся от последовательности тестируемой мертвой направляющей последовательности, и сравнении связывания или скорости расщепления реакции последовательности-мишени для тестируемой и контрольной направляющей последовательностей. Другие способы анализа также возможны и будут понятны квалифицированному специалисту в данной области. Мертвая последовательность направляющей молекулы может быть выбрана так, чтобы она была нацелена на любую последовательность-мишень. В некоторых вариантах осуществления изобретения последовательность-мишень является последовательностью в геноме клетки.
[466] Как объяснено в настоящем описании ниже, несколько структурных параметров обеспечивают надлежащий остов для таких мертвых направляющих молекул. Мертвые направляющие последовательности направляющей, как правило, короче, чем соответствующие последовательности направляющей молекулы, что приводит к активному расщеплению РНК. В конкретных вариантах осуществления изобретения мертвые направляющие молекулы на 5%, 10%, 20%, 30%, 40%, 50% короче, чем соответствующие направляющие молекулы с той же мишенью.
[467] Как объяснено ниже и известно в данной области, одним аспектом специфичности нацеливания на РНК посредством гРНК является последовательность прямого повтора, которая должна быть надлежащим образом связаны с такими направляющими молекулами. В частности, это подразумевает, что последовательности прямых повторов конструируют в соответствии с природой нацеленного на РНК фермента. Таким образом, имеющиеся структурные данные для подтвержденных мертвых направляющих последовательностей могут использоваться для разработки определенных эквивалентов C2c2. Структурное подобие, наблюдаемое, например, для ортологичного домена нуклеазы HEPN и двух или более эффекторных белков C2c2 может использоваться для конструирования эквивалентов мертвых направляющих молекул. Таким образом, длина и последовательность направляющей мертвой молекулы, описанной в настоящем описании, могут быть надлежащим образом изменены для отражения таких определенных эквивалентов C2c2, позволяя образование комплекса CRISPR и успешное связывание с РНК-мишенью, в то же время не допуская продуктивной нуклеазной активности.
[468] Использование мертвых направляющих молекул в контексте настоящего изобретение, а также состояние уровня техники, обеспечивает удивительную и неожиданную платформу для биологии сетей и/или системной биологии и в применениях in vitro, ex vivo и in vivo, делая возможным мультиплексированное нацеливание на гены, и в особенности двунаправленное мультиплексированное нацеливание на гены. До применения мертвых направляющих молекул направленное изменение множественных мишеней было затруднительным и в некоторых случаях невозможным. С использованием мертвых направляющих молекул становятся возможными множественные мишени, и, как следствие, множественные активности, например, в отдельной клетке, в отдельном животном или в отдельном пациенте. Такое мультиплексирование может быть единовременным или разделенным необходимыми промежутками времени.
[469] Например, мертвые направляющие молекулы позволяют использовать гРНК в качестве инструмента нацеливания на гены без сопутствующей нуклеазной активности, в то же время обеспечивая направленный инструмент активации или репрессии. Направляющая РНК, включающая мертвую направляющую молекулу, может быть изменена, чтобы в дополнительно включать элементы таким образом, чтобы допускать активацию или репрессию активности гена, в частности адаптерный белок (например, аптамеры), как описано в настоящем описании, обеспечивая функциональное размещение эффекторов генов (например, активаторы или репрессоры активности гена). Одним из примеров является включение аптамеров, как объяснено в настоящем описании и известно из уровня техники. Путем конструирования такой гРНК, включающей мертвую направляющую молекулу для встраивания взаимодействующих с белком аптамеров (Konermann et al., "Genome-scale transcription activation by an engineered CKISPR-Cas9 complex," doi:10.1038/nature14136, включенная в настоящее описание в качестве ссылки), можно добиться сборки множественных различающихся эффекторных доменов. Ее также можно моделировать на основе природных процессов.
[470] Таким образом, одним аспектом является гРНК по изобретению, включающая мертвую направляющую молекулу, причем направляющая РНК далее включает модификации, которые обеспечивают активацию или репрессию генов, как описано в настоящем описании. Мертвая гРНК может включать один или более аптамеров. Аптамеры могут быть специфичными к генным эффекторам, генным активаторам или генным репрессорам. Альтернативно этому, аптамеры могут быть специфичными к белку, который в свою очередь специфичен и привлекает/связывает определенный генный эффектор, генный активатор или генный репрессор. В случае множественных участков для привлечения активатора или репрессора, предпочтительно, чтобы участки были специфичны либо к активаторам, либо к репрессорам. В случае множественных участков связывания активатора или репрессора, такие участки могут быть специфичными к одним и тем же активаторам или одним и тем же репрессорам. Такие участки могут также быть специфичными к различным активаторам или различным репрессорам. Эффекторы, активаторы, репрессоры могут присутствовать в форме слитых белков.
[471] В одном из аспектов изобретение относится к способу селекции последовательности мертвой направляющей молекулы нацеливания на РНК для направления функционализированной CRISPR системы к локусу в организме, включающему: a) определение местонахождения одного или более мотивов CRISPR в гене локуса; b) анализ последовательность длиной 20 нуклеотидов в нисходящем направлении каждого мотива CRISPR путем: i) определения содержания GC-пар в последовательности; и ii) определения наличия нецелевых соответствий в первых 15 нуклеотидах последовательности в геноме организма; c) отбора последовательности для использования в гРНК, если содержание GC-пар в последовательности составляет 70% или менее и нецелевые соответствия не идентифицированы. В одном варианте осуществления последовательность отбирается, если содержание GC-пар составляет 50% или менее. В одном варианте осуществления последовательность отбирается, если содержание GC-пар составляет 40% или менее. В одном варианте осуществления последовательность отбирается, если содержание GC-пар составляет 30% или менее. В одном варианте осуществления анализируют две или более последовательностей и отбирают последовательность, имеющую самое низкое содержание GC-пар. В одном варианте осуществления нецелевые соответствия обнаруживаются в регуляторных последовательностях организма. В одном варианте осуществления изобретения такой локус является регуляторным регионом. Один аспект обеспечивает мертвую гРНК, включающую нацеливающую последовательность, отобранную согласно вышеупомянутым способам.
[472] В одном из аспектов изобретение относится к мертвой направляющей РНК для нацеливания функционализированной системы CRISPR на локус гена в организме. В одном варианте осуществления изобретения мертвая гРНК включает нацеливающую последовательность, где содержание GC-пар в последовательности-мишени составляет 70% или менее, и первые 15 нуклеотидов нацеливающей последовательности не соответствуют последовательности-мишени в нисходящем направлении от мотива CRISPR в регуляторной последовательности другого локуса в организме. В некоторых вариантах осуществления изобретения содержание GC-пар в нацеливающей последовательности составляет 60% или менее, 55% или менее, 50% или менее, 45% или менее, 40% или менее, 35% или менее или 30% или менее. В некоторых вариантах осуществления изобретения содержание GC-пар в нацеливающей последовательности составляет от 70% до 60%, от 60% до 50%, от 50% до 40% или от 40% до 30%. В одном варианте осуществления изобретения нацеливающая последовательность имеет самое низкое содержание GC-пар среди потенциальных нацеливающих на локус последовательностей.
[473] В одном варианте осуществления изобретения первые 15 нуклеотидов мертвой направляющей молекулы соответствуют последовательности-мишени. В другом варианте осуществления изобретения первые 14 нуклеотидов мертвой направляющей молекула соответствуют последовательности-мишени. В другом варианте осуществления изобретения первые 13 нуклеотидов мертвой направляющей молекулы соответствуют последовательности-мишени. В другом варианте осуществления изобретения первые 12 нуклеотидов мертвой направляющей молекулы соответствуют последовательности-мишени. В другом варианте осуществления изобретения первые 11 нуклеотидов мертвой направляющей молекулы соответствуют последовательности-мишени. В другом варианте осуществления изобретения первые 10 нуклеотидов мертвой направляющей молекулы соответствуют последовательности-мишени. В одном варианте осуществления изобретения первые 15 нуклеотидов мертвой направляющей молекулы не соответствуют последовательности-мишени в нисходящем направлении от мотива CRISPR в регуляторной области другого локуса. В других вариантах осуществления изобретения первые 14 нуклеотидов, первые 13 нуклеотидов мертвой направляющей молекулы, первые 12 нуклеотидов направляющей молекула, первые 11 нуклеотидов мертвой направляющей молекула или первые 10 нуклеотидов мертвой направляющей молекулы не соответствуют последовательности-мишени в нисходящем направлении от мотива CRISPR в регуляторной области другого локуса. В других вариантах осуществления изобретения первые 15 нуклеотидов, 14 нуклеотидов, 13 нуклеотидов, 12 нуклеотидов или 11 нуклеотидов мертвой направляющей молекулы не соответствуют последовательности-мишени в нисходящем направлении от мотива CRISPR в геноме.
[474] В определенных вариантах осуществления изобретения мертвая направляющая РНК включает дополнительные нуклеотиды на 3'-конце, не соответствующие последовательности-мишени. Таким образом, мертвая направляющая РНК, которая включает первые 20-28 нуклеотидов в нисходящем направлении от мотива CRISPR, может быть удлинена на 3'-конце.
Общие положения
[475] В одном из аспектов изобретение относится к системе связывания нуклеиновой кислоты. Гибридизация РНК in situ с комплементарными зондами является технологией с большими возможностями. Обычно, чтобы обнаружить нуклеиновые кислоты путем гибридизации, используются флуоресцентные олигонуклеотиды ДНК. Увеличение эффективности удалось достигнуть определенными модификациями, такими как закрытые нуклеиновые кислоты (LNA), однако остается потребность в эффективных и универсальных альтернативах. Изобретение обеспечивает эффективную и приспосабливаемую систему для гибридизации in situ.
[476] В вариантах осуществления изобретения понятия "направляющая последовательность" и "направляющая РНК" используются взаимозаменяемо, как и в упоминаемых выше документах, таких как WO 2014/093622 (PCT/US 2013/074667). В широком смысле, направляющая последовательность представляет собой любую нуклеотидную последовательность, имеющую достаточную комплементарность с полинуклеотидной последовательностью-мишенью для гибридизации с последовательностью-мишенью и обеспечения специфичного к последовательности связывания комплекса CRISPR с последовательностью-мишенью. В некоторым вариантах осуществления, уровень комплементаности между направляющей последовательностью и соответствующей последовательностью-мишенью при условии оптимального выравнивания с использованием надлежащего алгоритма выравнивания составляет примерно или превышает 50%, 60%, 75%, 80%, 85%, 90%, 95%, 97,5%, 99% или более. Оптимальное выравнивание может быть определено с использованием любого подходящего алгоритма для выравнивания последовательностей, не ограничивающими примерами которого являются алгоритм Смита-Ватермана, алгоритм Нидлмана-Вунша, алгоритмы, основанные на преобразовании Барроуза-Виллера (например, Burrows Wheeler Aligner), ClustalW, Clustal X, BLAT, Novoalign (Novocraft Technologies; доступен на www.novocraft.com), ELAND (Illumina, San Diego, CA), SOAP (доступен на soap.genomics.org.cn) и Maq (доступен на maq.sourceforge.net). В некоторых вариантах осуществления направляющая последовательность имеет длину примерно или более 5, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 75 или более нуклеотидов. В некоторых вариантах осуществления, направляющая последовательность имеет длину менее чем примерно 75, 50, 45, 40, 35, 30, 25, 20, 15, 12 или менее нуклеотидов. Предпочтительно, чтобы направляющая последовательность имела длину 10-30 нуклеотидов. Способность направляющей последовательности направлять специфичное к последовательности связывание комплекса CRISPR с последовательностью-мишенью может быть количественно оценена любым подходящим способом анализа. Например, компоненты системы CRISPR, достаточные для образования комплекса CRISPR, включая тестируемую направляющую последовательность, могут быть доставлены в клетку, содержащую соответствующую последовательность-мишень, например, путем трансфекции векторами, кодирующими компоненты последовательности CRISPR, с последующей количественной оценкой сайтов преимущественного расщепления последовательности-мишени, в частности, анализом Surveyor, как описано в настоящем описании. Сходным образом, расщепление полинуклеотидной последовательности-мишени может быть оценено в пробирке, содержащей последовательность-мишень, компоненты комплекса CRISPR, включая тестируемую направляющую последовательность и контрольную направляющую последовательность, при сравнении связывания или скорости расщепления последовательности-мишени для реакций с тестовой и контрольной направляющими последовательностями. Другие способы анализа также возможны и будут понятны квалифицированному специалисту в данной области. В некоторых вариантах осуществления последовательность-мишень является последовательностью в геноме клетки. Иллюстративные последовательности-мишени включают те, которые являются уникальными в геноме-мишени.
[477] В целом и в описании настоящего изобретения понятие "вектор" обозначает молекулу нуклеиновой кислоты, способную транспортировать другую нуклеиновую кислоту, с которой она связана. Векторы включают, но не ограничиваются ими, молекулы нуклеиновых кислот, являющиеся одноцепочечными, двухцепочечными или частично двухцепочечными; нуклеиновые кислоты, которые включают один или более свободных концов, не имеют свободных концов (например, кольцевые); нуклеиновые кислоты, которые представляют собой ДНК, РНК или ту и другую и другие разновидности полинуклеотидов, известных в данной области. Один из типов векторов называется "плазмидой", что означает кольцевую двухцепочечную ДНК-петлю, в которую могут быть внесены дополнительные участки ДНК, в том числе стандартными способами молекулярного клонирования. Другим типом векторов является вирусный вектор, в котором находятся последовательности, полученные из вирусных ДНК или РНК в векторе для упаковки в вирус (например, ретровирусы, дефектные по репликации ретровирусы, аденовирусы, дефектные по репликации аденовирусы и аденоассоциированные вирусы). Вирусные векторы также включают полинуклеотиды, переносимые вирусом для трансфекции клетки хозяина. Некоторые векторы способны к автономной репликации в клетке-хозяине, в которую они введены (например, бактериальные векторы, имеющие бактериальный ориджин репликации и векторы эписом млекопитающих). Другие векторы (например, неэписомные векторы млекопитающих) встраиваются в геном клетки-хозяина при введении в клетку хозяина и благодаря этому реплицируются вместе с геномом хозяина. Более того, некоторые векторы способны направлять экспрессию генов, с которыми они непосредственно связаны. Такие векторы называются в настоящем описании "экспрессирующими векторами". Векторы для экспрессии и обеспечивающие экспрессию в эукариотической клетке могут называться в настоящем описании "эукариотическими экспрессирующими векторами". Часто используемыми векторами в способах рекомбинантных ДНК являются плазмиды.
[478] Рекомбинантные экспрессирующие векторы могут включать нуклеиновую кислоту по изобретению в форме, подходящей для экспрессии такой нуклеиновой кислоты в клетке-хозяине, что означает, что такие рекомбинантные экспрессирующие векторы содержат один или более регуляторных элементов, которые могут быть выбраны в соответствии с используемыми для экспрессии клетками хозяина и которые функционально связаны с последовательностью нуклеиновой кислоты, которая должна быть экспрессирована. В рекомбинантном векторе экспрессии "функционально связанный" должно означать, что нуклеотидная последовательность-мишень связана с регуляторным(и) элементом(ами) таким образом, что экспрессия такой нуклеотидной последовательности является возможной (например, в системе транскрипции/трансляции in vitro или в клетке хозяина при введении вектора в клетку хозяина).
[479] Термин "регуляторный элемент" используется для обозначения промоторов, энхансеров, участков внутренней посадки рибосомы (IRES) и других элементов контроля экспрессии (например, сигналы терминации транскрипции, такие как сигналы полиаденилирования и поли(U)-последовательности). Такие регуляторные элементы описаны, например, в Goeddel, GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990). Регуляторные элементы включают те, что обеспечивают конститутивную экспрессию нуклеотидной последовательности в различных типах клеток, и те, что обеспечивают экспрессию нуклеотидной последовательности только в определенных клетках-хозяевах (например, тканеспецифические регуляторные последовательности). Тканеспецифический промотор может обеспечивать экспрессию преимущественно в желаемой целевой ткани, такой как мышца, нейрон, кость, кожа, кровь, конкретных органах (например, печень, поджелудочная железа) или конкретных типах клеток (например, лимфоцитах). Регуляторные элементы могут также управлять экспрессией в зависимости от времени, например в зависимости от стадии клеточного цикла или стадии развития, и при этом могут быть также специфичными к тканям или типу клеток. В некоторых вариантах осуществления изобретения вектор включает один или более промоторов РНК-полимеразы III (например, 1, 2, 3, 4, 5 или более промоторов РНК-полимеразы III), один или более промоторов РНК-полимеразы II (например, 1, 2, 3, 4, 5 или более промоторов РНК-полимеразы II), один или более промоторов РНК-полимеразы I (например, 1, 2, 3, 4, 5 или более промоторов РНК-полимеразы I) или их комбинацию. Примеры промоторов РНК-полимеразы III включают, но не ограничиваются ими, промоторы U6 и H1. Примеры промоторов РНК-полимеразы II включают, но не ограничиваются ими, промоторы вируса саркомы Рауса (RSV) (необязательно с энхансером RSV), промотор цитомегаловируса (CMV), (необязательно с энхансером CMV) [см., например, Boshart et al., Cell, 41:521-530 (1985)], промотор SV40, промотор дигидрофолатредуктазы, промотор p-актина, промотор фосфоглицеролкиназы (PGK) и промотор EF1a. Термин "регуляторный элемент" относится и к элементам-энхансерам, таким как WPRE; энхансеры CMV; R-U5'-сегмент в LTR HTLV-I (Mol. Cell. Biol., Vol. 8(1), p. 466-472, 1988); энхансер SV40 и последовательность интрона между 2 и 3 экзонами P-глобина кролика (Proc. Natl, Acad. Sci. USA., Vol. 78(3), p. 1527-31, 1981). Квалифицированный специалист в данной области оценит, что конструкция вектора экспрессии может зависеть от таких факторов как выбранная клетка-хозяин, трансфекция которой будет производиться, желаемый уровень экспрессии и т.д. Вектор может быть введен в клетки таким образом, чтобы получались транскрипты, белки или пептиды, включая слитые белки или пептиды, кодируемые нуклеиновыми кислотами, как описано в настоящем описании (например, транскрипты системы коротких палиндромных повторов, регулярно расположенных кластерами (CRISPR), их белки, ферменты, мутантные формы, слитые белки и т.д.)
[480] Преимущественные векторы включают лентивирусы и аденоассоциированные вирусы, и типы таких векторов могут также быть выбраны для нацеливания на конкретные типы клеток.
[481] Как используют в настоящем описании, термин "cr-РНК" или "направляющая РНК" или "единственная направляющая РНК" или "sg-РНК" или "один или более компонентов нуклеиновых кислот" эффекторного белка локуса типа V или типа VI системы CRISPR Cas включает любую полинуклеотидную последовательность, имеющую достаточную комплементарность с последовательностью нуклеиновой кислоты-мишени для гибридизации с последовательностью нуклеиновой кислоты-мишени и прямого, специфичного к последовательности связывания комплекса, нацеленного на нуклеиновую кислоту, с последовательностью нуклеиновой кислоты-мишени. В некоторых вариантах осуществления изобретения степень комплементарности при оптимальном выравнивании с использованием подходящего алгоритма выравнивания приблизительно равна 50%, 60%, 75%, 80%, 85%, 90%, 95%, 97,5%, 99% или более. Оптимальное выравнивание может быть проведено с использованием любого подходящего алгоритма выравнивания последовательностей, примеры которого включают, но не ограничиваются ими алгоритм Смита-Ватермана, алгоритм Нидлмана-Вунша, алгоритмы, основанные на преобразовании Барроуза - Уилера (например, Burrows Wheeler Aligner), ClustalW, Clustal X, BLAST, Novoalign (Novocraft Technologies; доступен на www.novocraft.com), ELAND (Illumina, Сан-Диего, Калифорния, США), SOAP (доступен на soap.genomics.org.cn) и Maq (доступен на maq.sourceforge.net). Способность направляющей последовательности (в пределах направляющей РНК, нацеливающей на нуклеиновые кислоты) управлять прямым специфичным к последовательности связыванием комплекса, нацеленного на нуклеиновые кислоты, может быть оценена с помощью любого подходящего способа анализа. Например, компоненты системы CRISPR, нацеленной на нуклеиновые кислоты, достаточные для формирования комплекса, нацеленного на нуклеиновую кислоту, включая направляющую последовательность, подлежащую тестированию, могут быть предоставлены клетке-хозяину, имеющей соответствующую последовательность нуклеиновой кислоты-мишени, например, путем трансфекции векторами, кодирующими компоненты комплекса, нацеленного на нуклеиновую кислоту, с последующей оценкой предпочтительного нацеливания (например, расщепления) в последовательности нуклеиновой кислоты-мишени, например, в анализе Survey, описанном в настоящем описании. Аналогично расщепление последовательности нуклеиновой кислоты-мишени может быть оценено в пробирке путем представления клетке последовательности нуклеиновой кислоты-мишени, компонентов комплекса, нацеленного на нуклеиновые кислоты, включая направляющую последовательность, подлежащую тестированию, и контрольную направляющую последовательность, отличающаяся от тестируемой направляющей последовательности, и сравнения связывания или уровня расщепления последовательности-мишени между реакциями тестовой и контрольной направляющими последовательностями. Другие способы анализа возможны и понятны специалисту в данной области. Направляющая последовательность, и, следовательно, направляющая РНК, нацеливающая на нуклеиновые кислоты, могут быть выбраны для нацеливания на любую последовательность нуклеиновой кислоты-мишени. Последовательность-мишень может быть представлена ДНК. Последовательность-мишень может быть представлена любой последовательностью РНК. В некоторых вариантах осуществления изобретения последовательность-мишень может представлять собой последовательность в молекуле РНК, выбранной из группы, состоящей из матричной РНК (мРНК), пре-мРНК, рибосомной РНК (рРНК), транспортной РНК (тРНК), микроРНК, малой интерферирующей РНК (миРНК), малой ядерной РНК (мяРНК), малой ядрышковой РНК (мякРНК), двухцепочечной РНК (дцРНК), некодирующей РНК (нкРНК), длинной некодирующей РНК (днкРНК) и малой цитоплазматической РНК (мцРНК). В некоторых предпочтительных вариантах осуществления изобретения последовательность-мишень может представлять собой последовательность в молекуле РНК, выбранную из группы, состоящей из мРНК, пре-мРНК и рРНК. В некоторых предпочтительных вариантах осуществления изобретения последовательность-мишень может представлять собой последовательность в молекуле РНК, выбранную из группы, состоящей из нкРНК и днкРНК. В некоторых более предпочтительных вариантах осуществления изобретения последовательность-мишень может представлять собой последовательность в молекуле мРНК или молекуле пре-мРНК.
[482] В некоторых вариантах осуществления изобретения направляющая РНК, нацеленная на нуклеиновые кислоты, выбрана таким образом, чтобы вторичная структура направляющей РНК, нацеленной на РНК, была более компактной. В некоторых вариантах осуществления изобретения приблизительно или меньше чем приблизительно 75%, 50%, 40%, 30%, 25%, 20%, 15%, 10%, 5%, 1%, или менее нуклеотидов направляющей РНК, нацеленной на нуклеиновую кислоту, участвует в самокомплементарном спаривании оснований при оптимальном фолдинге. Оптимальный фолдинг может быть определен любым подходящим алгоритмом выявления фолдинга полинуклеотида. Некоторые программы основаны на вычислении минимальной свободной энергии Гиббса. Примером одного такого алгоритма является mFold, описанный Zuker и Stiegler (Nucleic Acids Res. 9 (1981), 133-148). В качестве примера другого алгоритма фолдинга можно привести онлайн вебсервер RNAfold, разработанный в Институте Теоретической Химии в Венском университете, который используют центроидный алгоритм предсказания структуры (см., например, A.R. Gruber et al, 2008, Cell 106(1): 23-24; и PA Carr and GM Church, 2009, Nature Biotechnology 27(12): 1151-62).
[483] В определенных вариантах осуществления изобретения направляющая РНК или cr-РНК может включать, по существу состоять из или состоять из последовательности прямого повтора (DR) и последовательности направляющей молекулы или последовательности спейсера. В некоторых вариантах осуществления изобретения направляющая РНК или cr-РНК может включать, по существу состоять из или состоять из последовательности прямого повтора (DR), слитой или связанной с последовательностью направляющей молекулы или последовательностью спейсера. В некоторых вариантах осуществления изобретения последовательность прямого повтора (DR) может быть расположена в восходящем направлении (т.е. 5') от последовательности направляющей молекулы или последовательности спейсера. В других вариантах осуществления изобретения последовательность прямого повтора может быть расположена в нисходящем направлении (т.е. 3') от последовательности направляющей молекулы или последовательности спейсера.
[484] В определенных вариантах осуществления изобретения cr-РНК включает шпилечную структуру, предпочтительно единственную шпилечную структуру. В некоторых вариантах осуществления изобретения шпилечная структура, предпочтительно единственная шпилечная структура, формирует последовательность прямого повтора.
[485] В определенных вариантах осуществления изобретения длина спейсера направляющей РНК составляет от 15 до 35 нуклеотидов. В определенных вариантах осуществления изобретения длина спейсера направляющей РНК составляет по меньшей мере 15 нуклеотидов, предпочтительно по меньшей мере 18 нуклеотидов, например, по меньшей мере 19, 20, 21, 22 или более нуклеотидов. В некоторых вариантах осуществления изобретения длина спейсера составляет от 15 до 17 нуклеотидов, например, 15, 16 или 17 нуклеотидов, от 17 до 20 нуклеотидов, например, 17, 18, 19 или 20 нуклеотидов, от 20 до 24 нуклеотидов, например, 20, 21, 22, 23 или 24 нуклеотидов, от 23 до 25 нуклеотидов, например, 23, 24 или 25 нуклеотидов, от 24 до 27 нуклеотидов, например, 24, 25, 26 или 27 нуклеотидов, от 27-30 нуклеотидов, например, 27, 28, 29 или 30 нуклеотидов, 30-35 нуклеотидов, например, 30, 31, 32, 33, 34 или 35 нуклеотидов или 35 нуклеотидов или более.
[486] Заявители также провели пробный эксперимент для проверки способности белков типа V, таких как C2c1 или C2c3, к нацеливанию и расщеплению ДНК. Этот эксперимент во многом повторяет подобную работу для гетерологичной экспрессии StCas9 в E. coli (Sapranauskas, R. et al. Nucleic Acids Res 39, 9275-9282 (2011)). Заявители ввели плазмиду, содержащую одновременно PAM и ген устойчивости к антибиотикам, в гетерологичный штамм E. coli, и затем поместили на среду, содержащую соответствующий антибиотик. В случае расщепления ДНК плазмиды заявители не наблюдали жизнеспособных колоний.
[487] Более подробно анализ ДНК-мишени может быть проведен следующим образом. В анализе используются два штамма E.coli. Один из штаммов имеет плазмиду, которая кодирует локус эндогенного эффекторного белка бактериального штамма. Другой штамм имеет пустую плазмиду (например, pACYC184, контрольный штамм). Все возможные PAM длиной 7 или 8 п.н. представлены на плазмиде устойчивости к антибиотикам (pUC19 с геном устойчивости к ампициллину). PAM расположена рядом с последовательностью протоспейсера 1 (ДНК-мишень для первого спейсера в локусе эндогенного эффекторного белка). Были клонированы две библиотеки PAM. Одна библиотека содержит последовательности 5'-конца протоспейсера, состоящие из 8 случайных п.н. (например, всего 65 536 различных PAM=комплексность). Другая библиотека содержит последовательности 3'-конца протоспейсера, состоящие из 7 случайных п.н. (например, полная комплексность - 16 384 различных PAM). Обе библиотеки были клонированы, чтобы иметь в среднем 500 плазмид, содержащих каждый возможный PAM. Тестовый и контрольный штаммы были трансформированы библиотеками 5'-PAM и 3'-PAM в ходе отдельных трансформаций, трансформированные клетки были помещены в культуру на среду, содержавшую ампициллин. Распознавание и последующее разрезание/интерференция посредством плазмиды делают клетку неустойчивой к ампициллину и прекращают ее рост. Приблизительно через 12 ч после трансформации все колонии, образованных тестовым и контрольным штаммами, были собраны и из них была выделена плазмидная ДНК. Плазмидная ДНК использовалась в качестве матрицы для ПЦР-амплификации и последующего глубокого секвенирования. Представленность всех PAM в нетрансформированных библиотеках показала ожидаемую представленность PAM в трансформированных клетках. Представленность всех PAM, найденных в штаммах контроля, показала фактическую представленность. Представленность всех PAM в тестовом штамме показала, какие PAM не распознаются ферментом. Сравнение с контрольным штаммом позволяет извлекать последовательности с обедненным PAM.
[488] Для минимизации токсичности и неспецифических эффектов важно управлять концентрацией доставленной направляющей РНК. Оптимальные концентрации направляющей РНК, нацеленной на нуклеиновую кислоту, могут быть определены путем тестирования различных концентраций в клеточных моделях или эукариотических моделях животных, не являющихся человеком, глубокое секвенирование может быть использовано для анализа степени модификации в потенциальных геномных локусах, не являющихся мишенями. Концентрация для доставки in vivo должна быть подобрана так, чтобы обеспечивать наибольший уровень целевой модификации мишени при минимальном уровне нецелевой модификации. Предпочтительным является получение системы нацеливания на нуклеиновые кислоты из системы CRISPR типа VI. В некоторых вариантах осуществления изобретения один или более элементов системы нацеливания на нуклеиновые кислоты получены из конкретного организма, включающего эндогенную систему нацеливания на РНК. В конкретных вариантах осуществления изобретения фермент Cas типа VI, нацеленный на РНК, представляет собой C2c2. Примеры белков Cas включают, но не ограничиваются ими, Cas1, Cas1B, Cas2, Cas3, Cas4, Cas5, Cas6, Cas7, Cas8, Cas9 (также известный как Csn1 и Csx12), Cas10, Csy1, Csy2, Csy3, Cse1, Cse2, Csc1, Csc2, Csa5, Csn2, Csm2, Csm3, Csm4, Csm5, Csm6, Cmr1, Cmr3, Cmr4, Cmr5, Cmr6, Csb1, Csb2, Csb3, Csx17, Csx14, Csx10, Csx16, CsaX, Csx3, Csx1, Csx15, Csf1, Csf2, Csf3, Csf4, их гомологи или модифицированные версии. В вариантах осуществления изобретения белок типа VI, такой как C2c2, упомянутый в настоящем описании, также включает гомологи или ортологи белков типа VI, таких как C2c2. Термины "ортолог" (также обозначаемый в настоящем описании как "ortologue") и "гомолог" (также обозначаемый в настоящем описании как "homologue") хорошо известны в данной области. Посредством дальнейшего руководства "гомолог" белка как используют в настоящем описании, представляет собой белок того же самого вида, который выполняет ту же самую или подобную функцию, что и белок, гомологом которого он является. Гомологичные белки могут иметь сходную или частично сходную структуру, но это не обязательно. "Ортолог" белка, как используют в настоящем описании, является белком другого вида, который выполняет ту же самую или подобную функцию, что и белок, ортологом которого он является. Ортологичные белки могут иметь сходную или частично сходную структуру, но это не обязательно. В конкретных вариантах осуществления изобретения гомологи или ортологи белков типа VI, таких как C2c2, упомянутый в настоящем описании, имеют гомологию или идентичность последовательностей по меньшей мере 80%, более предпочтительно по меньшей мере 85%, еще более предпочтительно по меньшей мере 90%, например, по меньшей мере 95% с белками типа VI, такими как C2c2. В следующих вариантах осуществления изобретения гомологи или ортологи белков типа VI, таких как C2c2, упомянутый в настоящем описании, имеют идентичность последовательности по меньшей мере 80%, более предпочтительно по меньшей мере 85%, еще более предпочтительно по меньшей мере 90%, например, по меньшей мере 95% с белками типа VI дикого типа, такими как C2c2.
[489] В варианте осуществления изобретения белки Cas типа VI, нацеленные на РНК, могут быть ортологами C2c2 организмов из родов, которые включают, но не ограничены следующими: Corynebacter, Sutterella, Legionella, Treponema, Filifactor, Eubacterium, Streptococcus, Lactobacillus, Mycoplasma, Bacteroides, Flaviivola, Flavobacterium, Sphaerochaeta, Azospirillum, Gluconacetobacter, Neisseria, Roseburia, Parvibaculum, Staphylococcus, Nitratifractor, Mycoplasma и Campylobacter. Вид организма, принадлежащего к данным родам, может быть обсужден в данной заявке иным образом.
[490] Некоторые способы идентификации ортологов ферментов системы CRISPR-Cas могут включать идентификацию tracr-последовательности в геномах-мишенях. Идентификация tracr-последовательностей может быть связана со следующими шагами: поиск прямых повторов или последовательности tracr-помощника в базе данных, чтобы идентифицировать область CRISPR, включающую фермент CRISPR; поиск гомологичных последовательностей в области CRISPR, фланкирующем фермент CRISPR в смысловом и антисмысловом направлениях; поиск терминаторов транскрипции и вторичных структур; идентификация любой последовательности, которая не является прямым повтором или последовательностью tracr-помощника, но имеет больше чем 50% идентичность с прямым повтором или последовательностью tracr-помощника как потенциальная tracr-последовательность; поиск последовательностей терминатора транскрипции, ассоциированных с потенциальной tracr-последовательностью.
[491] Следует принимать во внимание, что любой из функциональных видов активности, описанных в настоящем описании, может быть встроена способами инженерии в ферменты CRISPR из других ортологов, включая химерные ферменты, включающие фрагменты многих ортологов. Примеры таких ортологов описаны в настоящем описании. Таким образом, химерные ферменты могут включать фрагменты ортологов фермента CRISPR организма, который включает, но не ограничен следующими: Corynebacter, Sutterella, Legionella, Treponema, Filifactor, Eubacterium, Streptococcus, Lactobacillus, Mycoplasma, Bacteroides, Flaviivola, Flavobacterium, Sphaerochaeta, Azospirillum, Gluconacetobacter, Neisseria, Roseburia, Parvibaculum, Staphylococcus, Nitratifractor, Mycoplasma и Campylobacter. Химерный фермент может включать первый фрагмент и второй фрагмент, фрагменты могут происходить из ортологов фермента CRISPR организмов, принадлежащих к родам, упомянутым в настоящем описании, или видам, упомянутым в настоящем описании; предпочтительно фрагменты происходят из ортологов фермента CRISPR различных видов.
[492] В вариантах осуществления изобретения, эффекторный белок типа VI, нацеленный на РНК, в частности, белок C2c2, упоминаемый в настоящем описании, также охватывает функциональный вариант C2c2 или его гомолог, или ортолог. "Функциональный вариант" белка, как используют в настоящем описании, относится к варианту такого белка, который сохраняет, по меньшей мере частично, активность этого белка. Функциональные варианты могут включать мутанты (которые могут иметь инсерции, делеции или замены), включая полиморфы, и т.д. Также в функциональные варианты включены продукты слияния такого белка с другим, обычно неродственным, белком, нуклеиновой кислотой, полипептидом или пептидом. Функциональные варианты могут быть естественными или искусственными. Предпочтительные варианты осуществления изобретения могут включать не встречающийся в природе или сконструированный способами инженерии эффекторный белок типа VI, нацеленный на РНК, например, C2c1/C2c3, или его ортолог или гомолог.
[493] В варианте осуществления изобретения кодон молекулы (молекул) нуклеиновой кислоты, кодирующей эффекторный белок типа VI, нацеленный на РНК, в частности, C2c2, или его ортолог или гомолог, может быть оптимизирован для экспрессии в эукариотической клетке. Эукариотический организм может быть таким, как описано в настоящем описании. Молекула (молекулы) нуклеиновой кислоты может быть сконструирована способами инженерии или не встречаться в природе.
[494] В варианте осуществления изобретения эффекторный белок типа VI, нацеленный на РНК, в частности, C2c2 (или его ортолог или гомолог), может включать одну или более мутаций (следовательно, молекула (молекулы) нуклеиновой кислоты, кодирующая этот белок, имеет ту же самую мутацию (мутации)). Мутации могут быть внесены искусственно и могут включать, но не ограничиваться ими, одну или более мутациий в каталитическом домене. Примеры каталитических доменов в отношении фермента Cas9 могут включать, но не ограничиваться ими, домены RuvC I, RuvC II, RuvC III и HNH.
[495] В варианте осуществления изобретения белок Cas типа VI, такой как C2c2, или его ортолог или гомолог, может включать одну или более мутаций. Мутации могут быть внсены искусственно и могут включать, но не ограничиваться ими, одну или более мутаций в каталитическом домене. Примеры каталитических доменов в отношении фермента Cas могут включать, но не ограничиваться ими, домены RuvC I, RuvC II, RuvC III, HNH и домены HEPN.
[496] В одном варианте осуществления изобретения белок Cas типа VI, такой как C2c2, или его ортолог или гомолог, может использоваться в качестве универсального белка, связывающего нуклеиновые кислоты, в слиянии или будучи функционально связанным с функциональным доменом. Иллюстративные функциональные домены могут включать, но не ограничиваются ими, инициатор трансляции, активатор трансляции, репрессор трансляции, нуклеазы, в частности, рибонуклеазы, сплайсосомы, магнитные гранулы, индуцируемый/контролируемй светом домен или химически индуцируемый/контролируемый домен.
[497] В некоторых вариантах осуществления изобретения ферментативная активность неизмененного эффекторного белка, нацеленного на нуклеиновые кислоты, заключается в расщеплении. В некоторых вариантах осуществления изобретения эффекторный белок нацеливания на РНК может направлять расщепление одной или обеих нуклеиновых кислот (ДНК или РНК) цепи в местоположении около последовательности-мишени, таком как в пределах последовательности-мишени и/или в пределах последовательности, комплементарной последовательности-мишени, или в последовательностях, ассоциированных с последовательностью-мишенью. В некоторых вариантах осуществления изобретения белок Cas, нацеленный на нуклеиновую кислоту, может направлять расщепление одной или обеих цепей ДНК или РНК в пределах приблизительно 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 50, 100, 200, 500 или более пар оснований от первого или последнего нуклеотида последовательности-мишени. В некоторых вариантах осуществления изобретения расщепление может быть тупым, т.е. производить тупые концы. В некоторых вариантах осуществления изобретения расщепление может быть ступенчатым, т.е. производить липкие концы. В некоторых вариантах осуществления изобретения расщепление может быть ступенчатым с 5'-выступающими концами, например, 5'-выступающими концами длиной 1-5 нуклеотидов. В некоторых вариантах осуществления изобретения расщепление может быть ступенчатым с 3'-выступающими концами, например, 3'-выступающими концами длиной 1-5 нуклеотидов. В некоторых вариантах осуществления изобретения вектор кодирует белок Cas, нацеленный на нуклеиновые кислоты, который может быть мутантным по сравнению с соответствующим ферментом дикого типа, таким образом, что мутантный белок Cas, нацеленный на нуклеиновую кислоту, имеет пониженную способность расщеплять одну или обе цепи ДНК или РНК полинуклеотида-мишени, содержащего последовательность-мишень. В качестве еще одного примера, мутации могут быть внесены в два или более каталитических домена белка Cas (RuvC I, RuvC II и RuvC III или домен HNH или домен HEPN), чтобы мутантный белок Cas практически утратил активность расщепления РНК. Как описано в настоящем описании, мутации могут быть внесены в соответствующие каталитические домены эффекторного белка C2c2, чтобы активность расщепления ДНК эффекторного белка C2c2 была недостаточной или существенно уменьшенной. В некоторых вариантах осуществления изобретения эффекторный белок, нацеленный на нуклеиновую кислоте, считается имеющий существенный недостаток активности расщепления РНК, когда активность расщепления РНК мутантного фермента составляет не больше чем 25%, 10%, 5%, 1%, 0,1%, 0,01% или менее активности расщепления нуклеиновой кислоты немутантной формы фермента; например, когда активность расщепления нуклеиновой кислоты мутантной формы равна нулю или незначительна по сравнению с немутантной формой. Эффекторный белок может быть идентифицирован в отношении общего класса ферментов, которые являются гомологичными самой большой нуклеазе со множественными нуклеазными доменами системы CRISPR типа V/типа VI. Наиболее предпочтительно использование эффекторного белка типа V/типа VI, такого как C2c2. Под "происходящим" заявители подразумевают, что происходящий фермент в значительной степени основан, в смысле наличия высокой степени гомологии последовательности, на ферменте дикого типа, но что он некоторым образом мутирован (модифицирован), как известно в данной области или как описано в настоящем описании.
[498] Снова, следует принимать во внимание, что термины "Cas и фермент CRISPR", "белок CRISPR" и "белок Cas" обычно используются взаимозаменяемо и во всех случаях упоминания в настоящем описании относится относятся по аналогии к новым эффекторным белкам CRISPR, далее описанными в настоящем описании, если иное не очевидно, например, при конкретной отсылке к Cas9. Как упомянуто выше, многие нумерации остатков, используемые в настоящем описании, относятся к эффекторному белку локуса CRISPR типа V/типа VI. Однако следует принимать во внимание, что это изобретение включает еще множество эффекторных белков из других видов микроорганизмов. В некоторых вариантах осуществления изобретения, Cas может конститутивно присутствовать, или индуцируемо присутствовать, или кондиционально присутствовать, или быть введенным, или быть доставленным. Оптимизация Cas может использоваться для усиления функций или разработки новых функций, могут быть получены химерные белки Cas. Cas может использоваться в качестве универсального белка, связывающего нуклеиновые кислоты.
[499] Как правило, в контексте эндогенной системы, нацеленной на нуклеиновую кислоту, образование комплекса, нацеленного на нуклеиновую кислоту (включающего направляющую РНК, гибридизованную с последовательностью-мишенью, и образующую комплекс с одним или более нацеленными на нуклеиновую кислоту эффекторными белками) приводит к расщеплению одной или обеих цепей ДНК или РНК в пределах или рядом (например, в 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 50, или более пар оснований от) с последовательностью-мишенью. Как используют в настоящем описании, термин "последовательность (последовательности), ассоциированная с локусом-мишень", относится к последовательностям вблизи последовательности-мишени (например, в пределах 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 50 или более пар оснований от последовательности-мишени, где последовательность-мишень находится в локусе-мишени).
[500] Примером кодон-оптимизированной последовательности в данном случае является последовательность, оптимизированная для экспрессии в эукариотическом организме, например, в человеке (т.е. являющаяся оптимизированной для экспрессии в человеке) или другом эукариотическом организме, животном или млекопитающем, как описано в настоящем описании; см., например, кодон-оптимизированную для человека последовательность SaCas9 в WO 2014/093622 (PCT/US2G13/074667) в качестве примера кодон-оптимизированной последовательности (как это известно из уровня техники и настоящего описания, кодон оптимизированная кодирующая молекула(ы) нуклеиновой(ых) кислот(ы), особенно в случае эффекторного белка (например, C2c2), входит в компетенцию квалифицированного специалиста). В то время как именно представленное выше описание является предпочтительным, будет также понятно, что другие примеры являются возможными и известна оптимизация кодонов для видов хозяев, отличных от человека, или оптимизация кодонов для определенных органов. В некоторых вариантах осуществления кодирующая фермент последовательность, в которой закодирован нацеленный на ДНК/РНК белок Cas, является кодон-оптимизированной для экспрессии в определенных клетках, в частности, эукариотических клетках. Такие эукариотические клетки конкретного организма, или могут происходить из него, который может быть млекопитающим, включая, но не ограничиваясь этим человека, отличного от человека эукариотического организма, животного или млекопитающего, как описано в настоящем описании, например, мышь, крысу, кролика, собаку, домашний скот или отличного от человека млекопитающего или примата. В некоторых вариантах осуществления процессы модификации генетической принадлежности эмбриональной линии клеток человеческих индивидов и/или процессы модификации генетической принадлежности эмбриональной линии клеток животных, которые с большой долей вероятности могут принести им страдания без получения существенной пользы для медицинских приложений для человека или животных, а также получаемые при этих процессах животные, могут быть исключены. В целом, под оптимизацией кодонов понимается процесс изменения последовательности нуклеиновой кислоты для усиления экспрессии в клетках-мишенях хозяина путем замены по меньшей мере одного кодона (например, примерно или более чем 1, 2, 3, 4, 5, 10, 15, 20, 25, 50 или более кодонов) нативной последовательности кодонами, которые более часто или наиболее часто используются в генах этой клетки-хозяина при сохранении нативной аминокислотной последовательности. Различные виды демонстрируют определенное предпочтение для определенных кодонов конкретных аминокислот. Предпочтение кодонов (различия в использовании кодонов между организмами) часто коррелирует с эффективностью трансляции матричных РНК (мРНК), что в свою очередь считается зависящим от, в числе прочего, от свойств транслируемых кодонов или доступности молекул определенной транспортной РНК (тРНК). Преобладание определенных тРНК обычно отражает наиболее часто используемые при синтезе пептидов кодоны. В соответствии с этим, гены могут быть адаптированы для оптимальной экспрессии генов в конкретном организме на основе оптимизации кодонов. Таблицы частот использования кодонов свободно доступны, например, в базе "Codon Usage Database", доступной на www.kazusa.oijp/codon/, причем такие таблицы могут быть адаптированы различными способами. См. Nakamura, Y. et al. "Codon usage tabulated from the international DNA sequence databases: status for the year 2000" Nucl. Acids Res, 28:292 (2000). Также доступны компьютерные алгоритмы для оптимизации кодонов для конкретной последовательности для экспрессии в определенной клетке-хозяине, в частности, такие как Gene Forge (Aptagen; Джакобус, Пенсильвания, США). В некоторых вариантах осуществления один или более кодонов (например, 1, 2, 3, 4, 5, 10, 15, 20, 25, 50, более или все кодоны) в последовательности, кодирующей нацеленный на ДНК/РНК белок Cas соответствуют наиболее часто используемым кодонам для конкретной аминокислоты.
[501] В некоторых вариантах осуществления вектор кодирует нацеленный на нуклеиновую кислоту эффекторный белок, такой как нацеленный на РНК эффекторный белок типа V, в частности C2c2, либо ортолог или гомолог такового, содержащий одну или более последовательностей сигнала ядерной локализации (NLS), к примеру примерно или более 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS. В некоторых вариантах осуществления такой нацеленный на РНК эффекторный белок содержит примерно или более чем 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS на N-конце или вблизи него, примерно или более чем 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более NLS на C-конце или вблизи него или их сочетание (например, ноль или по меньшей мере один или более NLS на N-конце или более NLS на C-конце). В случае наличия более одной NLS, каждая из них может быть отобрана независимо от других, в частности, отдельная NLS может присутствовать в виде более чем одной копии и/или в сочетании с одной или более NLS, представленной одной или более копией. В некоторых вариантах осуществления NLS считается находящейся вблизи N- или C- конца, когда ближайшая аминокислота NLS находится на расстоянии менее примерно 1, 2, 3, 4, 5, 10, 15, 20, 25, 30, 40, 50 или более аминокислот полипептидной цепи от N-конца или C-конца. Неограничивающие примеры NLS включают последовательности NLS, полученные из: NLS большого T-антигена вируса SV40, имеющий аминкоислотную последовательность PKKKRKV; NLS нуклеоплазмина (например, двухкомпонентная NLS, имеющий последовательностью KRPAATKKAGQAKKKK); NLS c-myc, имеющий аминокислотную последовательность PAAKRVKLD или RQRRNELKRSP; NLS hRNPA1 M9, имеющий аминокислотную последовательность NQSSNFGPMKGGNFGGRSSGPYGGGGQYFAKPRNQGGY; последовательность RMRIZFKNKGKDTAELRRRRVEVSVELRKAKKDEQILKRRNV домена IBB импортина-альфа; последовательности VSRKRPRP и PPKKARED T-белка миомы; последовательность POPKKKPL p53 человека; последовательность SALIKKKKKMAP c-ab1 IV мыши; последовательности DRLRR и PKQKKRK вируса гриппа NS1; последовательность RKLKKKIKKL дельта-антигена вируса гепатита; последовательность REKKKFLKRR белка Mx1 мыши; последовательность KRKGDEVDGVDEVAKKKSKK поли(АДФ-рибоза)-полимеразы и последовательность RKCLQAGMNLEARKTKK глюкокортикоида рецепторов стероидных гормонов. В целом, одна или более NLS являются достаточными, чтобы вызвать накопление нацеленного на ДНК/РНК белка Cas в поддающихся обнаружению количествах в ядре эукариотической клетки. В целом, выраженность активности ядерной локализации может быть результатом деятельности ряда NLS в нацеленном на нуклеиновые кислоты эффекторном белке, использования определенных NLS или сочетания этих факторов. Отслеживание накопления в ядре может быть выполнено с помощью любого подходящего способа. Например, отслеживаемый маркер может быть слит с нацеленным на нуклеиновую кислоту белком, так что его расположение в клетке может быть визуализировано, в частности, в комбинации со способами определения расположения ядра (например, с помощью специфичного для ядра красителя, такого как DAPI). Ядра клеток могут также быть выделены из клеток, содержимое которых может затем быть проанализировано с использованием любого подходящего способа отслеживания белка, такого как иммунногистохимия, вестерн-блоттинг или анализ активности белка. Накопление в ядре может быть определено и косвенными способами, например, путем анализа эффекта образования нацеленного на нуклеиновые кислоты комплекса (например, изменения активности экспрессии при образовании нацеленного на ДНК или РНК комплекса и/или активности белка Cas по нацеливанию на РНК), при сравнении с контролем, не подвергнутым воздействию нацеленного на нуклеиновую кислоту белка Cas или нацеленного на нуклеиновую кислоту комплекса, или подвергнутым действию нацеленного на нуклеиновую кислоту белка Cas, лишенного одного или более NLS. В предпочитаемом варианте описываемые в настоящем описании комплексы и системы эффекторного белка C2c2 и комплексы и системы эффекторного белка C2c2 включают NLS, присоединенный к C-концу белка.
[502] В некоторых вариантах осуществления один или более векторов, управляющих экспрессией одного или более систем нацеливания на нуклеиновые кислоты вводят в клетку таким образом, что экспрессия элементов системы нацеливания на нуклеиновую кислоту управляет образованием нацеленного на нуклеиновую кислоту комплекса на одном или более участках-мишенях. Например, нацеленный на нуклеиновую кислоту эффекторный белок и нацеливающая на нуклеиновую кислоту направляющая РНК могут быть функционально связаны с отдельными регуляторными элементами в отдельных векторах. Молекула(ы) системы, нацеленной на нуклеиновую кислоту, может быть доставлена к трансгенныму эффекторному белку, нацеленному на нуклеиновую кислоту, являющемуся белком животного или млекопитающего, конститутивно, индуцибельно или кондиционально экспрессирующего эффекторный белок, нацеленный на нуклеиновую кислоту, или животного или млекопитающего, экспрессирующего эффекторный белок, нацеленный на нуклеиновую кислоту иным образом, или имеющего клетки, содержащие эффекторный белок, нацеленный на нуклеиновую кислоту, находящийся там вследствие предшествующего введения вектора или векторов, кодирующих и экспрессирующих in vivo эффекторный белок, нацеленный на нуклеиновую кислоту. Альтернативно этому, два или более элементов, экспрессированных при участии одного или различных регуляторных элементов, могут быть введены в единый вектор, с одним или более дополнительных векторов, обеспечивающих какие-либо компоненты системы, нацеленной на нуклеиновую кислоту, не включенных в первый вектор. Компоненты системы, нацеленной на нуклеиновую кислоту, объединенные в единый вектор, могут быть собраны в любой подходящей ориентации, например, в которой один элемент расположен в восходящем направлении относительно 5'-конца или в нисходящем направлении относительно 3'-конца второго элемента. Кодирующая последовательность одного элемента может быть расположена на той же или противоположной цепи кодирующей последовательности второго элемента и иметь одну и ту же или противоположную ориентацию. В некоторых вариантах осуществления один и тот же промотор контролирует экспрессию транскриптов, кодирующих эффекторный белок, нацеленный на нуклеиновую кислоту, и направляющую РНК, нацеленную на нуклеиновую кислоту, встроенных в одну или более последовательностей интронов (например, каждая в отдельном интроне, две или более по меньшей мере в одном интроне или все в одном и том же интроне). В некоторых вариантах осуществления эффекторный белок, нацеленный на нуклеиновую кислоту, и нацеливающая на нуклеиновую кислоту направляющая РНК могут быть функционально связаны с или экспрессироваться с одного и того же промотора. Средства доставки, векторы, частицы, наночастицы, составы и их компоненты для экспрессии одного или более элементов системы, нацеленной на нуклеиновую кислоту, используются в вышеупомянутых документах, в частности, в WO 2014/093622 (PCT/US2013/074667). В некоторых вариантах осуществления вектор содержит одну или более участков вставок, таких как последовательности узнавания эндонуклеаз рестрикции (также называемые "сайтом клонирования"). В некоторых вариантах осуществления один или более участков вставок (например, примерно или более чем 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более участков вставок) расположены в восходящем и/или нисходящем направлении относительно одной или более элементов последовательностей одного или более векторов. В некоторых вариантах осуществления вектор включает два или более участков вставок, что делает возможным введение в качестве вставок направляющих последовательностей в каждый участок. При такой организации две или более направляющих последовательностей могут включать две или более копий единичной направляющей последовательности, две или более различных направляющих последовательностей или их комбинацию. При использовании множества различных направляющих последовательностей единственная конструкция экспрессии может быть использована для нацеливания активности связывания нуклеиновых кислот на множественно различных соответствующих последовательностей-мишеней в клетке. К примеру, единственный вектор может включать примерно или более 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20 или более направляющих последовательностей. В некоторых вариантах осуществления, может быть предоставлено примерно или более 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более таких содержащих направляющую последовательность векторов, и необязательно они также могут быть доставлены в клетку. В некоторых вариантах осуществления вектор содержит регуляторный элемент, функционально связанный с кодирующей фермент последовательностью. Эффекторный белок, нацеленный на нуклеиновую кислоту или направляющая РНК или направляющие РНК, нацеливающие на нуклеиновую кислоту, могут быть доставлены независимо; предпочтительно, чтобы по меньшей мере один(одна) из них был(а) доставлен(а) посредством комплекса частиц или наночастиц. мРНК эффекторного белка, нацеленного на нуклеиновую кислоту, может быть доставлена прежде направляющей РНК, нацеливающей на нуклеиновую кислоту, для того, чтобы обеспечить время для экспрессии эффекторного белка, нацеленного на нуклеиновую кислоту. мРНК эффекторного белка, нацеленного на нуклеиновую кислоту, может быть введена на 1-12 часов (предпочтительно примерно 2-6 часов) раньше введения направляющей РНК, нацеливающей на нуклеиновую кислоту. Альтернативно этому, мРНК нацеленного на нуклеиновую кислоту эффекторного белка и направляющая РНК, нацеливающая на нуклеиновую кислоту, могут быть введены одновременно. Предпочтительно, когда вторая усиливающая доза направляющей РНК вводится на протяжении 1-12 часов (предпочтительно примерно 2-6 часов) после начального введения мРНК нацеленного нуклеиновую кислоту эффекторного белка+направляющей РНК. Дополнительные введения нацеленного на нуклеиновую кислоту эффекторного белка могут оказаться полезными, чтобы достичь наиболее эффективных уровней модификации генома и/или транскриптома.
[503] В одном аспекте изобретение относится к способам использования одного или более компонентов системы нацеливания на нуклеиновую кислоту. Такой нацеленный на нуклеиновую кислоту комплекс по изобретению обеспечивает эффективный инструмент модификации ДНК- или РНК-мишени, являющейся одно- или двухцепочечной, линейной или суперспирализованной. Такой нацеленный на нуклеиновую кислоту комплекс по изобретению имеет разнообразные преимущества, включая модификацию (например, удаление, вставка, перемещение, инактивация, активация) ДНК- или РНК-мишени в множестве типов клеток. Как таковой нацеленный на нуклеиновую кислоту комплекс по изобретению имеет широкий спектр применений, например, в генной терапии, фармакологическом скрининге, диагностике заболеваний и их прогнозировании. Иллюстративный нацеленный на нуклеиновую кислоту комплекс включает нацеленный на ДНК или РНК эффекторный белок в комплексе с направляющей РНК, гибридизованной с последовательностью-мишенью в представляющем интерес локусе-мишени.
[504] В одном из вариантов осуществления настоящее изобретение относится к способу расщепления РНК-мишени. Данный способ может включать модификацию РНК-мишени с использованием нацеленного на нуклеиновую кислоту комплекса, который связывает РНК-мишень и осуществляет расщепление указанной РНК-мишени. В одном из вариантов осуществления нацеленный на нуклеиновую кислоту комплекс по изобретению при введении в клетку может вносить разрыв (например, одноцепочечный или двухцепочечный разрыв) в последовательность РНК. Например, такой способ может быть использован для расщепления связанной с заболеванием РНК в клетке. Например, экзогенная РНК-матрица, включающая последовательность, которую необходимо встроить, фланкированная вышележащей последовательностью и нижележащей последовательностью, может быть введена в клетку. Такие вышележащие и нижележащие последовательности характеризуются сходством последовательности с обоими концами участка встраивания в РНК. При желании, донорная РНК может представлять собой мРНК. Экзогенная РНК-матрица содержит последовательность, которую необходимо встроить (например, мутированную РНК). Последовательность для встраивания может представлять собой последовательность, эндогенную или экзогенную для клетки. Примерами последовательностей для встраивания являются кодирующая белок РНК или некодирующая РНК (например, микроРНК). Таким образом, последовательность для встраивания может быть функционально связана с надлежащей последовательностью или последовательностями контроля. Альтернативно, встраиваемая последовательность может выполнять регуляторную функцию. Вышележащие и нижележащие последовательности в экзогенной РНК выбирают так, чтобы стимулировать рекомбинацию между РНК-мишенью и донорной РНК. Вышележащая последовательность является последовательностью РНК, имеющей сходство последовательности с последовательностью РНК в восходящем направлении от целевого участка интеграции. Сходным образом, нижележащая последовательность является последовательностью РНК, имеющей сходство последовательности с последовательностью РНК в нисходящем направлении от целевого участка интеграции. Вышележащая и нижележащая последовательности в экзогенной РНК-матрице могут иметь 75%, 80%, 85%, 90%, 95% или 100% идентичность последовательности с последовательностью РНК-мишени. Предпочтительно, чтобы вышележащая и нижележащая последовательности в экзогенной РНК-матрице имели примерно 95%, 96%, 97%, 98%, 99% или 100% идентичность последовательности с последовательностью РНК-мишени. В некоторых способах вышележащая и нижележащая последовательности экзогенной РНК-матрицы имеют 99% или 100% идентичность последовательности с последовательности РНК-мишени. Вышележащие или нижележащие последовательности могут содержать от примерно 20 п.н. до примерно 2500 п.н., например, примерно 50, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 1600, 1700, 1800, 1900, 2000, 2100, 2200, 2300, 2400 или 2500 п.н. В некоторых способах иллюстративная вышележащая или нижележащая последовательность имеет от примерно 200 п.н. до примерно 2000 п.н., от примерно 600 п.н. до примерно 1000 п.н. или более конкретно от примерно 700 п.н. до примерно 1000 п.н. В некоторых способах экзогенная РНК-матрица может дополнительно содержать маркер. Такой маркер может облегчить скрининг целевых встраиваний. Примеры подходящих маркеров включают участки рестрикции, флуоресцентные белки или метки, по которым возможен отбор. Экзогенная РНК-матрица по изобретению может быть получена с помощью рекомбинантных способов (см., например, Sambrook et al., 2001 и Ausubel et al., 1996). Согласно способу модификации РНК-мишени путем встраивания экзогенной РНК-мишени разрыв (например, двухцепочечный или одноцепочечный разрыва в одноцепочечной или двухцепочечной ДНК или РНК) вносится в последовательность ДНК или РНК нацеленным на нуклеиновую кислоту комплексом, причем такой разрыв репарируется за счет гомологичной рекомбинации с экзогенной РНК-матрицы таким образом, что матрица оказывается встроенной в РНК-мишень. Присутствие двухцепочечного разрыва облегчает встраивание матрицы. В других вариантах осуществления настоящее изобретение относится к способу модификации экспрессии РНК в эукариотической клетке. Такой способ включает увеличение или уменьшение экспрессии полинуклеотида-мишени путем использования нацеленного на нуклеиновую кислоту комплекса, связывающего ДНК или РНК (например, мРНК или пре-мРНК). В некоторых способах РНК-мишень может быть инактивирована для того, чтобы влиять на модификацию экспрессии в клетке. Например, при связывании нацеленного на РНК комплекса с последовательностью-мишенью в клетке РНК-мишень инактивируется так, что такая последовательность не транслируется, кодируемый ей белок не образуется или такая последовательность не функционирует так, как последовательность дикого типа. Например, белок, или транскрипты микроРНК или пре-микроРНК не образуются. РНК-мишень нацеленного на РНК комплекса может представлять собой любую РНК, эндогенную или экзогенную для данной эукариотической клетки. Например, РНК-мишень может представлять собой РНК, находящуюся в ядре эукариотической клетки. РНК-мишень может представлять собой последовательность (например, мРНК или пре-мРНК), кодирующую генный продукт (например, белок) или некодирующую последовательность (например, нкРНК, lnc-РНК, тРНК или рРНК). Примеры РНК-мишени включают последовательность, ассоциированную с сигнальными биохимическими путями, например, ассоциированную с сигнальными биохимическими путями РНК. Примеры РНК-мишени включают РНК, ассоциированные с заболеваниями. "Ассоциированные с заболеваниями" РНК обозначает любые РНК, используемые для синтеза продуктов трансляции на аномальном уровне или в аномальной форме в клетках, полученных из затронутых заболеванием тканей в сравнении с тканями или клетками не затронутого заболеванием контроля. Это может быть РНК, транскрибированная с гена, которая экспрессируется на аномально высоком уровне; это может быть РНК, транскрибированная с гена, который экспрессируется на аномально низком уровне, причем измененная экспрессия коррелирует с появлением и/или прогрессированием заболевания. Ассоциированные с заболеванием РНК также упоминают как РНК, транскрибированные с гена, несущего мутацию(и) или генетическую вариацию, которые напрямую ответственны или связаны с неравновесностью сцепления с геном(ами), который(ые) вовлечен в этиологию заболевания. Продукт трансляции может быть известен или не известен и его количество может иметь нормальный или аномальный уровень. РНК-мишень нацеленного на РНК комплекса может представлять собой любую РНК, эндогенную или экзогенную для такой эукариотической клетки. Например, РНК-мишень нацеленного на РНК комплекса может представлять собой РНК, находящуюся в ядре эукариотической клетки. Такая РНК-мишень может быть последовательностью (например, мРНК или пре-мРНК), кодирующей продукт гена (например, белок) или некодирующей последовательностью (например, нкРНК, lnc-РНК, тРНК или рРНК).
[505] В некоторых вариантах осуществления изобретения способ может позволять нацеленному на нуклеиновую кислоту комплексу связываться с ДНК- или РНК-мишенью, чтобы произвести расщепление указанной ДНК- или РНК-мишени, таким образом, изменяя ДНК- или РНК-мишень, где нацеленный на нуклеиновые кислоты комплекс включает нацеленный на нуклеиновые кислоты эффекторный белок в комплексе с направляющей РНК, гибридизованной с последовательностью-мишенью в указанной ДНК- или РНК-мишени. В одном аспекте изобретение относится к способу модификации экспрессии ДНК или РНК в эукариотической клетке. В некоторых вариантах осуществления изобретения способ позволяет нацеленному на нуклеиновые кислоты комплексу связываться с ДНК или РНК, таким образом, что указанное связывание приводит к увеличению или уменьшению экспрессии указанной ДНК или РНК; где нацеленный на нуклеиновые кислоты комплекс включает нацеленный на нуклеиновые кислоты эффекторный белок в комплексе с направляющей РНК. Применимы соображения и условия, сходные с теми, что указаны выше для способов модификации ДНК- или РНК-мишени. В действительности, варианты взятия образцов, культивирования и повторного введения применяются во многих аспектах настоящего изобретения. В одном аспекте изобретение относится к способам модификации ДНК- или РНК-мишени в эукариотической клетке, которая может происходить in vivo, ex vivo или in vitro. В некоторых вариантах осуществления изобретения способ включает взятие клетки или популяции клеток у человека или животного, не являющегося человеком, и модификацию этой клетки или клеток. Культивирование ex vivo может происходить на любой стадии. Клетка или клетки могут быть повторно введены в животное, не являющееся человеком, или растение. Для повторно введенных клеток особо предпочтительно, чтобы эти клетки являлись стволовыми клетками.
[506] Действительно, в любом аспекте изобретения нацеленный на нуклеиновую кислоту комплекс может включать нацеленный на нуклеиновую кислоту эффекторный белок в комплексе с направляющей РНК, которая гибридизованной с последовательностью-мишенью.
[507] Изобретение относится к инженерии и оптимизации систем, способов и композиций, используемых для контроля экспрессии генов, включающим нацеливание на последовательности ДНК и РНК, которые связаны с нацеленной на нуклеиновые кислоты системой и ее компонентами. В предпочтительных вариантах осуществления изобретения эффекторным белком (ферментом) является белок типа VI, такой как C2c2. Преимущество способов по настоящему изобретению состоит в том, что система CRISPR минимизирует или избегает неспецифического связывания и его побочных эффектов. Эта система достигается с использованием систем, организованных так, что они имеют высокую степень специфичности к ДНК- или РНК-мишени.
[508] Что касается нацеленного на нуклеиновую кислоту комплекса или системы, предпочтительно, чтобы tracr-последовательность имела одну или более шпилек и составляла 30 или более нуклеотидов в длину, 40 или более нуклеотидов в длину или 50 или более нуклеотидов в длину; длина последовательности cr-РНК варьировалась между 10 и 30 нуклеотидами, а нацеленный на нуклеиновую кислоту эффекторный белок принадлежал к эффекторным белкам типа VI.
[509] В некоторых вариантах осуществления изобретения эффекторный белок может представлять собой C2c2p Listeria sp., предпочтительно C2c2p Listeria seeligeria, более предпочтительно C2c2p Listeria seeligeria серовара 1/2b штамма SLCC3954, и последовательность cr-РНК может составлять 44-47 нуклеотидов в длину с прямыми повторами (DR) на 5'-конце длиной 29 нуклеотидов и спейсером длиной от 15 нуклеотидов до 18 нуклеотидов.
[510] В некоторых вариантах осуществления изобретения эффекторный белок может представлять собой C2c2p Leptotrichia sp., предпочтительно C2c2p Leptotrichia shahii, более предпочтительно C2c2p Leptotrichia shahii DSM 19757, и последовательность cr-РНК может составлять 42-58 нуклеотидов в длину с прямым повтором на 5'-конце длиной по меньшей мере 24 нуклеотидов, таким как прямой повтор (DR) на 5'-конце длиной 24-28 нуклеотидов, и спейсер длиной по меньшей мере 14 нуклеотидов, такой как от 14 нуклеотидов до 28 нуклеотидов, или по меньшей мере 18 нуклеотидов, такой как 19, 20, 21, 22 или более нуклеотидов, такой как 18-28, 19-28, 20-28, 21-28 или 22-28 нуклеотидов.
[511] В некоторых вариантах осуществления изобретения эффекторный белок может относиться к эффекторным белкам локусов типа VI, более конкретно C2c2p, последовательность cr-РНК может иметь длину 36-63 нуклеотидов, предпочтительно от 37 нуклеотидов до 62 нуклеотидов или от 38 нуклеотидов до 61 нуклеотидов или от 39 нуклеотидов до 60 нуклеотидов, более предпочтительно от 40 нуклеотидов до 59 нуклеотидов или от 41 нуклеотидов до 58 нуклеотидов, наиболее предпочтительно от 42 нуклеотидов до 57 нуклеотидов. Например, cr-РНК может включать, по существу состоять из или состоять из прямого повтора (DR), предпочтительно на 5'-конце длиной от 26 нуклеотидов до 31 нуклеотидов, предпочтительно от 27 нуклеотидов до 30 нуклеотидов, еще более предпочтительно 28 нуклеотидов или 29 нуклеотидов в длину или по меньшей мере 28 или 29 нуклеотидов в длину, и спейсера длиной от 10 нуклеотидов до 32 нуклеотидов, предпочтительно от 11 нуклеотидов до 31 нуклеотидов, более предпочтительно от 12 нуклеотидов до 30 нуклеотидов, еще более предпочтительно от 13 нуклеотидов до 29 нуклеотидов, и наиболее предпочтительно от 14 нуклеотидов до 28 нуклеотидов, например 18-28 нуклеотидов, 19-28 нуклеотидов, 20-28 нуклеотидов, 21-28 нуклеотидов или 22-28 нуклеотидов.
[512] В некоторых вариантах осуществления изобретения эффекторный белок может относиться к эффекторным белкам локусов типа VI, более конкретно C2c2p, и последовательность tracr-РНК может иметь длину по меньшей мере 60 нуклеотидов, такую как по меньшей мере 65 нуклеотидов или по меньшей мере 70 нуклеотидов, такую как от 60 нуклеотидов до 70 нуклеотидов, или от 60 нуклеотидов до 70 нуклеотидов, или от 70 нуклеотидов до 80 нуклеотидов, или от 80 нуклеотидов до 90 нуклеотидов, или от 90 нуклеотидов до 100 нуклеотидов, или от 100 нуклеотидов до 110 нуклеотидов, или от 110 нуклеотидов до 120 нуклеотидов, или от 120 нуклеотидов до 130 нуклеотидов, или от 130 нуклеотидов до 140 нуклеотидов, или от 140 нуклеотидов до 150 нуклеотидов, или больше, чем 150 нуклеотидов. См. иллюстративные примеры на фиг. 22-37.
[513] В определенных вариантах осуществления изобретения эффекторный белок может относиться к эффекторным белкам локусов типа VI, более конкретно C2c2p, поэтому tracr-РНК для расщепления может не требоваться.
[514] Использование двух различных аптамеров (каждый ассоциирован с отдельной нацеливающей на нуклеиновую кислоту направляющей РНК) позволяет использование слитых слитой конструкции активатор-адаптерный белок и слитых конструкций репрессор-адаптерный белок с различными нацеливающими на нуклеиновые кислоты направляющими РНК, для активации экспрессии только одной ДНК или РНК, подавляя другую. Их, наряду с их различными направляющими РНК можно вводить совместно, или по существу совместно, в мультиплексном подходе. Большое количество таких модифицированных нацеленных на нуклеиновые кислоты направляющих РНК может использоваться одновременно, например 10 или 20 или 30 и т.д., в то время как только одна (или по меньшей мере минимальное число) молекула эффекторного белка должна быть доставлена, так как сравнительно небольшое количество молекул эффекторного белка может использоваться с большим количеством модифицированных направляющих молекул. Адаптерный белок может быть ассоциирован (предпочтительно связан или слит с) одним или более активаторами или одним или более репрессорами. Например, адаптерный белок может быть ассоциирован с первым и вторым активаторами. Первый и второй активаторы могут быть одинаковыми, но предпочтительно они являются различными активаторами. Можно использовать три и более или даже четыре и более активатора (или репрессора), однако допустимый размер упаковки ограничивает их число 5 различными функциональными доменами. Использование линкеров является предпочтительным над прямым слиянием с адаптерным белком, где два или больше функциональных домена ассоциированы с адаптерным белком. Подходящие линкеры могут включать линкер GlySer.
[515] Также предусматривается, что нацеленный на нуклеиновые кислоты комплекс эффекторного белка и направляющей РНК в целом может быть ассоциирован с двумя или более функциональными доменами. Например, может быть два или более функциональных домена, связанных с нацеленным на нуклеиновые кислоты эффекторным белком, или может быть два или более функциональных домена, связанных с направляющей РНК (через один или более адаптерных белков), или может быть один или более функциональных доменов, связанных с нацеленным на нуклеиновые кислоты эффекторным белком, и один или более функциональных доменов, связанных с направляющей РНК (посредством одного или более адаптерных белков).
[516] Слитая конструкция адаптерного белка и активатора или репрессора может включать линкер. Например, могут использоваться линкеры GlySer GGGS. Они могут использоваться в повторах по 3 ((GGGGS)3) или 6, 9 или даже 12 или более, чтобы обеспечить подходящие длину, как требуется. Линкеры могут использоваться между направляющими РНК и функциональным доменом (активатор или репрессор), или между нацеленным на нуклеиновые кислоты эффекторным белком и функциональным доменом (активатор или репрессор). Линкеры используют для придания белку необходимой "механической гибкости".
[517] Изобретение охватывает нацеленный на нуклеиновую кислоту комплекс, включающий нацеленный на нуклеиновую кислоту эффекторный белок и направляющую РНК, где нацеленный на нуклеиновую кислоту эффекторный белок включает по меньшей мере одну мутацию, такую, что у нацеленного на нуклеиновую кислоту белка Cas есть не более 5% активности нацеленного на нуклеиновую кислоту белка Cas, не имеющего по меньшей мере одной мутации и необязательно по меньшей мере одну или более последовательностей ядерной локализации; направляющая РНК включает последовательность направляющей молекулы, способную к гибридизации с последовательностью-мишенью в представляющей интерес РНК в клетке; и где нацеленный на нуклеиновую кислоту эффекторный белок ассоциирован с двумя или более функциональными доменами, или по меньшей мере одна шпилечная структура направляющей РНК изменена вставкой отдельной последовательности (последовательностей) РНК, которая связана с одним или более адаптерными белками, и где адаптерный белок связан с двумя или более функциональными доменами; или нацеленный на нуклеиновую кислоту эффекторный белок ассоциирован с одним или более функциональными доменами, и по меньшей мере одна шпилечная структура направляющей РНК изменена вставкой отдельной последовательности (последовательностей) РНК, которая связана с одним или более адаптерными белками, и где адаптерный белок связан с одним или более функциональными доменами.
Общая характеристика доставки
Комплексы эффекторного белка C2c2 могут доставлять функциональные эффекторы
[518] В отличие от опосредованного CRISPR-Cas генного нокаута, который устраняет экспрессию перманентно, внося в ген мутации на уровне ДНК, нокдаун CRISPR-Cas позволяет временно сократить экспрессию генов с помощью искусственной транскрипции или факторов трансляции. Мутация ключевых остатков в доменах расщепления как ДНК, так и РНК, в белке C2c2 приводит к созданию каталитически неактивного C2c2. Каталитически неактивный C2c2 образует комплекс с направляющей РНК и локализуется в последовательности ДНК или РНК, определяемой доменом нацеливания этой направляющей РНК, однако, он не расщепляет ДНК- или РНК-мишень. Слияние неактивного белка C2c2 с эффекторным доменом, например, доменом репрессии транскрипции или трансляции, позволяет привлекать эффектор к любому участку ДНК или РНК, определяемому направляющей РНК. В определенных вариантах осуществления изобретения C2c2 может быть слит с доменом репрессии транскрипции и привлечен в область промотора гена. В частности, для генной репрессии предусматривается, что сайта связывания эндогенного фактора транскрипции может снизить экспрессию генов. В другом варианте осуществления изобретения неактивный C2c2 может быть слит с белком модификации хроматина. Изменение состояния хроматина может привести к уменьшению экспрессии гена-мишени. В следующих вариантах осуществления изобретения C2c2 может быть слит с доменом репрессии трансляции.
[519] В одном варианте осуществления изобретения молекулу направляющей РНК можно нацеливать на известные элементы ответа транскрипции (например, промоторы, энхансеры и т.д.), известные вышележащие активирующие последовательности и/или последовательности неизвестной или известной функции, которые могут иметь способность управлять экспрессией ДНК-мишени.
[520] В некоторых способах полинуклеотид-мишень может быть инактивирован для модификации экспрессии в клетке. Например, после связывания комплекса CRISPR с последовательностью-мишенью в клетке, целевой полинуклеотид инактивируется таким образом, что последовательность не транскрибируется, закодированный белок не синтезируется, или последовательность не функционирует, в отличие от последовательности дикого типа. Например, последовательности, кодирующие белок или микроРНК, могут быть инактивированы таким образом, что белок не синтезируется. В некоторых способах полинуклеотид-мишень может быть инактивирован, чтобы произвести модификацию экспрессии в клетке. Например, после связывания CRISPR комплекса с последовательностью РНК-мишени в клетке, полинуклеотид-мишень инактивируется таким образом, что последовательность не транслируется, влияя на уровень экспрессии белка в клетке.
[521] В конкретных вариантах осуществления изобретения фермент CRISPR содержит одну или более мутаций, отобранных из группы, состоящей из R597A, H602A, R1278A и H1283A, и/или одну или более мутаций, находящихся в домене HEPN фермента CRISPR, или иную мутацию, описанную в настоящем описании. В некоторых вариантах осуществления изобретения фермента CRISPR имеет одну или более мутаций в каталитическом домене, где во время транскрипции последовательность прямого повтора формирует единственную шпилечную структуру и направляющая последовательность руководит специфическим для последовательности связыванием комплекса CRISPR с последовательностью-мишенью, и где фермент далее включает функциональный домен. В некоторых вариантах осуществления изобретения функциональный домен представляет собой a. В некоторых вариантах осуществления изобретения функциональный домен представляет собой домен репрессии транскрипции, предпочтительно KRAB. В некоторых вариантах осуществления изобретения домен репрессии транскрипции представлен SID или конкатемерами SID (например, SID4X), В некоторых вариантах осуществления изобретения функциональный домен является эпигенетическим доменом модификации, обеспечивающим работу эпигенетического фермента модификации. В некоторых вариантах осуществления изобретения функциональный домен является доменом активации, который может быть доменом активации P65.
Доставка комплекса эффекторного белка C2c2 или его компонентов
[522] В настоящем описании, а также из уровня техники, предусматривается, что TALE-нуклеазы, системы CRISPR-Cas или их компоненты, или их молекулы нуклеиновых кислот, или молекулы нуклеиновых кислот, кодирующие или доставляющие их компоненты, могут быть доставлены системой доставки, описанной в настоящем описании как в общем виде, так и более подробно.
[523] Векторная доставка, например, плазмидная, вирусная доставка: фермент CRISPR, например белок типа V, такой как C2c2, и/или любая из РНК по настоящему изобретению, например направляющая РНК, могут быть доставлены с использованием любого подходящего вектора, например, плазмидных или вирусных векторов, таких как аденоассоциированный вирус (AAV), лентивирус, аденовирус или другие типы вирусных векторов или их комбинации. Эффекторные белки и одна или более направляющих РНК могут быть упакованы в один или более векторов, например, плазмидные или вирусные векторы. В некоторых вариантах осуществления изобретения вектор, например, плазмидный или вирусный вектор, доставляется в целевую ткань, например, путем внутримышечной инъекции, в то время как в других случаях доставка осуществляется внутривенно, трансдермально, интраназально, перорально, через слизистые оболочки или другими способами доставки. Такая доставка может быть в виде единичной дозы либо в виде нескольких доз. Специалисту в данной области понятно, что фактическая дозировка, которая должна быть доставлена, может значительно варьироваться в зависимости от ряда факторов, таких как выбор вектора, тип клетки-мишени, самого организма или ткани, общее состояние пациента, желаемая степень трансформации/модификации, способ введения, режим введения, тип желаемой трансформации/модификации и т.д.
[524] Такой препарат может в дальнейшем включать, например, носитель (в составе которого вода, водный раствор хлорида натрия, этанол, глицерин, лактоза, сахароза, фосфат кальция, желатин, декстран, агар, пектин, арахисовое масло, кунжутное масло и т.д.), растворитель, применяемый в фармакологии носитель (например, фосфатный солевой буфер), применяемый в фармакологии наполнитель и/или другие соединения, известные в данной области. Препарат, кроме того, может содержать одну или более применяемых в фармакологии солей, например, таких как соль неорганической кислоты, такая как гидрохлорид, гидробромид, фосфат, сульфат и т.д., и соли органических кислот, такие как ацетаты, пропионаты, малонаты, бензоаты и т.д. Кроме того, также использоваться могут вспомогательные вещества, такие как смачивающие или эмульгирующие, буферные растворы, гели или гелеобразователи, вкусовые добавки, красители, микросферы, полимеры, вещества, образующие суспензии и т.д. Кроме того, один или более других стандартно применяемых фармацевтических добавок, таких как консерванты, влагоудерживающие добавки, суспенизирующие добавки, поверхностно-активные добавки, антиоксиданты, противослеживающие добавки, наполнители, хелатирующие добавки, вещества оболочки, химические стабилизаторы и т.д. могут также быть использованы, особенно если дозировка имеет разбавленную форму. Подходящие иллюстративные компоненты включают микрокристаллическую целлюлозу, натрий карбокисметилцеллюлозу, полисорбат 80, фенилэтиловый спирт, хлорбутанол, сорбат калия, сорбиновую кислоту, двуокись серы, пропилгаллат, парабены, этилванилин, глицерин, фенол, парахлорофенол, желатин, альбумин и их комбинацию. Полное обсуждение применяемых в фармакологии наполнителей доступно в REMINGTON'S PHARMACEUTICAL SCIENCES (Mack Pub, Co., N.J. 1991), который включен в настоящее описание в качестве ссылки.
[525] В одном варианте осуществления изобретения, описанном в настоящем описании, доставка осуществляется с помощью аденовируса, который может быть доставлен в виде единичной вспомогательной дозы, содержащей по меньшей мере 1×105 частиц (также называемых единицами частиц, еч) аденовирусного вектора. В одном варианте осуществления изобретения настоящего изобетения, предпочтительная доза составляет по меньшей мере приблизительно 1×106 частиц (например, приблизительно 1×106-1×1012 частиц), более предпочтительно по меньшей мере приблизительно 1×107 частиц, более предпочтительно по меньшей мере приблизительно 1×108 частиц (например, приблизительно 1×108-1×1011 частиц или приблизительно 1×108-1×1012 частиц), и наиболее предпочтительно по меньшей мере приблизительно 1×109 частиц (например, приблизительно 1×109-1×1010 частиц или приблизительно 1×109-1×1012 частиц), или даже по меньшей мере приблизительно 1×1010 частиц (например, приблизительно 1×1010-1×1012 частиц) аденовирусного вектора. Альтернативно, доза включает не больше чем приблизительно 1×1014 частиц, предпочтительно не больше чем приблизительно 1×1013 частиц, еще более предпочтительно не больше чем приблизительно 1×1012 частиц, еще более предпочтительно не больше чем приблизительно 1×1011 частиц, и наиболее предпочтительно не больше чем приблизительно 1×1010 частиц (например, не больше чем приблизительно 1×109 частиц). Таким образом, доза может содержать единственную дозу аденовирусного вектора, например, с приблизительно 1×106 единиц частиц (еч), приблизительно 2×106 еч, приблизительно 4×106 еч, 1×107 еч, приблизительно 2×107 еч, приблизительно 4×107 еч, приблизительно 1×108 еч, приблизительно 2×108 еч, приблизительно 4×108 еч, приблизительно 1×109 еч, приблизительно 2×109 еч, приблизительно 4×109 еч, приблизительно 1×1010 еч, приблизительно 2×1010 еч, приблизительно 4×1010 еч, приблизительно 1×1011 еч, приблизительно 2×1011 еч, приблизительно 4×1011 еч, приблизительно 1×1012 еч, приблизительно 2×1012 еч или приблизительно 4×1012 еч аденовирусного вектора. См., например, аденовирусные векторы в заявке на патент США №8454972, B2 Nabel, et al., поданной 4 июня 2013 года, включенной в настоящее описание в качестве ссылки, и в ее колонке 29, строки 36-58. В одном варианте осуществления изобретения, описанном в настоящем описании, аденовирус доставляется посредством нескольких доз.
[526] В описанном в настоящем описании варианте осуществления изобретения доставка осуществляется через AAV. Терапевтически эффективная дозировка для in vivo доставки AAV человеку, как полагают, находится в диапазоне от приблизительно 20 приблизительно до 50 мл солевого раствора, содержащего от приблизительно 1×1010 до приблизительно 1×1010 раствора функционального AAV/мл. Дозировка может быть скорректирована для уравновешивания терапевтического эффекта и каких-либо побочных эффектов. В одном варианте осуществления изобретения, описанном в настоящем описнии, доза AAV обычно находится в следующем диапазоне концентраций: от приблизительно 1×105 до 1×1050 геномов AAV, от приблизительно 1×108 до 1×1020 геномов AAV, от приблизительно 1×1010 до приблизительно 1×1016 геномов или от приблизительно 1×1011 до приблизительно 1×1016 геномов AAV. Дозировка для человека может составлять приблизительно 1×1016 геномов AAV. В таких концентрациях можно доставить от приблизительно 0,001 мл до приблизительно 100 мл, от приблизительно 0,05 до приблизительно 50 мл или от приблизительно 10 до приблизительно 25 мл раствора-носителя. Другие эффективные дозировки могут быть легко определены квалифицированным специалистом в данной области посредством стандартных испытаний, на основе которых строится кривая зависимости ответа от дозы. См., например, заявку на патент США №8404658 B2 Hajjar, et al., поданную 26 марта 2013 года, колонка 27, строки 45-60.
[527] В одном варианте осуществления изобретения, описанном в настоящем описании, доставка производится через плазмиду. В случае таких плазмидных конструкций их дозировка должна быть достаточной для того, чтобы вызвать ответ. Например, подходящее количество ДНК-плазмиды в конструкциях плазмиды может составлять от приблизительно 0,1 до приблизительно 2 мг, или от приблизительно 1 пг до приблизительно 10 пг на человека массой 70 кг. Плазмиды по изобретению обычно включают (i) промотор: (ii) последовательность, кодирующую нацеленный на нуклеиновую кислоту фермент CRISPR, функционально связанный с указанным промотором; (iii) селективный маркер; (iv) ориджин репликации; и (v) терминатор транскрипции, находящийся ниже и функционально связанный с (ii). Плазмида может также кодировать РНК-компоненты комплекса CRISPR, но один или более из них может кодироваться другим вектором.
[528] Дозы, описанные в настоящем описании, основаны на средней массе человека, равной 70 кг. Определение частоты введения входит в компетенции практикующего медицинского или ветеринарного специалиста (например, врач, ветеринар) или ученого-специалиста в данной области. Также отмечается, что мыши, используемые в экспериментах, как правило, имеют массу приблизительно 20 г, и результаты экспериментов на мышах могут быть экстраполированы на индивида массой 70 кг.
[529] В некоторых вариантах осуществления изобретения молекулы РНК по изобретению доставляют в составе липосом или в составе, содержащим липофектин и т.п. и могут быть получены с помощью способов, известных специалисту в данной области. Такие способы описаны, например, в патентах США №5593972, 5589466 и 5580859, которые включены в настоящее описание в качестве ссылки в полном объеме. Системы доставки для на усиленной и улучшенной доставки миРНК в клетки млекопитающих были разработаны (см., например, Shen et al. FEBS Let. 2003, 539:111-114; Xia et al., Nat. Biotech. 2002, 20:1006-1010; Reich et al., Mol. Vision. 2003, 9: 210-216; Sorensen et al., J. Mol. Biol. 2003, 327: 761-766; Lewis et al., Nat. Gen. 2002, 32: 107-108 и Simeoni et al., NAR 2003, 31, 11: 2717-2724) и могут быть использованы в рамках настоящего изобретения. Недавно миРНК была успешно использована для ингибирования экспрессии генов у приматов (см., например, Tolentino et al., Retina 24(4):660) и может также быть применена в рамках настоящего изобретения.
[530] Действительно, РНК-доставка является эффективным способом для доставки in vivo. Возможно доставить нацеленный на нуклеиновую кислоту Cas, белок Cas9 и направляющую РНК - гРНК (и, например, матрицу для репарации HR) в клетки с использованием липосом или частиц. Таким образом, доставка нацеленного на нуклеиновую кислоту белка Cas/ фермента CRISPR, такого как Cas-белок Cas9 и/или доставка направляющих РНК по изобретения может производится в форме РНК с использованием микровезикул, липосом или частиц. Например, мРНК Cas и направляющая РНК могут быть упакованы в липосомальные частицы для доставки in vivo. Липосомальные реактивы для трансфекции, такие как липофектамин производства Life Technologies и другие реактивы, доступные на рынке, могут эффективно доставить молекулы РНК в печень.
[531] Предпочтительно, чтобы средства доставки РНК также включали доставку РНК через наночастицы (Cho, S, Goldberg, M, Son, S, Xu, Q, Yang, F, Mei, Y, Bogatyrev, S, Langer, R, and Anderson, D, Lipid-like nanoparticles for small interfering RNA delivery to endothelial cells, Advanced Functional Materials, 19; 3112-3118, 2010) или экзосомы (Schroeder, A, Levins, C, Cortez, C, Langer, R, and Anderson, D, Lipid-based nanotherapeutics for siRNA delivery, Journal of Internal Medicine, 267: 9-21, 2010, PMID: 20059641). Действительно, экзосомы, как показано, особенно полезны при доставке миРНК, системы, обладающей некоторым сходством с системой нацеливания на РНК. Например, El-Andaloussi S et al. ("Exosome-mediated delivery of siRNA in vitro and in vivo Nat Protoc. 2012 Dec;7(12):2112-26. doi: 10.1038/nprot.2012.131. Epub 2012 Nov 15.) описали, что экзосомы являются многообещающим инструментом доставки лекарственных средств через различные биологические барьеры и могут использоваться для доставки миРНК in vitro и in vivo. Данный подход состоит в производстве экзосом-мишеней посредством трансфекции вектора экспрессии, включающего экзосомный белок, слитый с лигандом-пептидом. Экзосомы далее проходят очистку и для отделения от клеточного супернатанта, далее РНК загружают в экзосомы. Доставка или введение согласно изобретению могут быть произведены с применением экзосом, к примеру, но не ограничиваясь этим, в мозг. Витамин Е (α-токоферол) может быть связан с нацеленным на нуклеиновую кислоту белком Cas и доставлен в мозг вместе с липопротеином высокой плотности (HDL), например, аналогично тому, как это было сделано Uno et al. (HUMAN GENE THERAPY 22:711-719 (June 2011)) для доставки милой интерферирующей РНК (миРНК)) в мозг. Мышам проводились вливание с применением осмотического микронасоса (модель 1007D; Alzet, Cupertino, Калифорния, США) фосфатного солевого буфера (PBS) или свободных TocsiBACE или Toc-siBACE/HDL и подсоединяли к набору для вливаний в мозг (Brain Infusion Kit 3, Alzet). Используемая для вливаний в мозг канюля помещалась примерно на 0,5 мм позади брегмы по центральной линии для вливания в третий дорзальный желудочек. Uno et al. установили, что не более 3 нмоль Toc-siRNA с HDL могут вызывать целевое уменьшение в степени, сравнимой с таковой при применении интракраниального способа введения (ICV). Сходная дозировка нацеленного на нуклеиновые кислоты эффекторного белка, конъюгированного с α-токоферолом и введенного совместно с нацеленным на мозг HDL, предполагается для человека в рамках настоящего изобретения, например, может предполагаться от примерно 3 нмоль до примерно 3 мкмоль нацеленного на нуклеиновую кислоту эффекторного белка, нацеленного в мозг. Zou et al. (HUMAN GENE THERAPY 22:465-475 (April 2011)) описывают способ опосредованной лентивирусом доставки кшРНК, нацеленной на PKCγ для подавления экспрессии генов in vivo в спинном мозге крыс. Zou et all. вводили примерно 10 мкл рекомбинантного лентивируса, имеющего титр 1×109 трансдуцирующих частиц (тч)/мл, через интратекальный катетер. Сходная дозировка нацеленного на нуклеиновую кислоту эффекторного белка, экспрессированного с лентивирусного вектора, нацеленного в мозг, также может предполагаться в рамках настоящего изобретения, например, также предполагается примерно 10-50 мл нацеленного на нуклеиновую кислоту эффекторного белка, направленного в мозг, с титром лентивируса 1×109 трансдуцирующих частиц (тч)/мл.
[532] В отношении локальной доставки в мозг, возможны различные ее пути. Например, материал может быть доставлен в стриатум, например, путем инъекции. Инъекция может быть произведена стереотактически при трепанации черепа.
[533] Повышение эффективности NHEJ или HR также является полезным для такой доставки. Предпочтительно, чтобы эффективность NHEJ была усилена одновременно экспрессируемыми ферментами процессинга концов, такими как Trex2 (Dumitrache et al. Genetics. 2011 August: 188(4): 787-797). Предпочтительно, чтобы эффективность HR была увеличена временным ингибированием машинерией NHEJ, в частности, Ku70 и Ku86. Эффективность HR может также быть увеличена путем одновременной экспрессии прокариотических или эукариотических ферментов рекомбинации, таких как RecBCD, RecA.
Общая характеристика Упаковки и Промоторов
[534] Способы упаковки молекул нуклеиновых кислот, кодирующих нацеленный на нуклеиновую кислоту эффекторный белок (такой как белок типа V, например C2c2), например, ДНК, в векторы, например, вирусные векторы, для опосредования модификации генома in vivo включают:
Для достижения опосредованного NHEJ нокаута гена:
Вектор на основе одного вируса:
Вектор, содержащий две или более кассет экспрессии:
Промотор-направляющая РНК-терминатор
Промотор-направляющая РНК (N-конец)-терминатор (до предела размера вектора)
Двойной вирусный вектор:
Вектор 1, содержащий одну экпрессирующую кассету для управления экспрессией нацеленного на нуклеиновую кислоту эффекторного белка (такого как белок типа V, например, C2c2)
Промотор-молекула нуклеиновой кислоты, кодирующая нацеленный на нуклеиновую кислоту эффекторный белок-терминатор
Вектор 2, содержащий одну или более экспрессирующих кассет для управления экспрессией одной или более направляющей(их) РНК
Промотор-направляющая РНК 1-терминатор
Промотор-направляющая РНК 1 (N-конец)-терминатор (до предела длины вектора)
Для опосредования гомологичной репарации.
В дополнение к подходам с одиночным и двойными векторами, дополнительный вектор используется для доставки матрицы репарации на основе гомологии.
[535] Промотор, используемый для управления экспрессией нуклеиновой кислоты, нацеленной на нуклеиновую кислоту эффекторным белком (таким как белок типа V, такой как C2c2) может включать:
ITR AAV может выступать в качестве промотора: это предпочтительно для того, чтобы избавиться от дополнительного промоторного элемента (который занимает место в векторе). Освобожденное в результате этого дополнительное пространство может быть использовано, чтобы управлять экспрессией дополнительных элементов (направляющая РНК и т.д.). Кроме того, активность ITR относительно слабее, так что он может использоваться для уменьшения потенциальной токсичности из-за сверхэкспрессии нацеленного на нуклеиновые кислоты эффекторного белка (такого как белок типа V, такой как C2c2).
Для повсеместной экспрессии можно использовать промоторы: CMV, CAG, CBh, PGK, SV40, тяжелых или легких цепей ферритина, и т.д.
Для экспрессии в мозге или другой экспрессии в ЦНС можно использовать промоторы: промотор синапсина I для всех нейронов, CaMKIIalpha для возбуждающих нейронов, GAD67, GAD65 или VGAT для GABA-ергических нейронов, и т.д.
Для экспрессии в печени можно использовать промотор альбумина.
Для экспрессии в легких можно использовать SP-B.
Для эндотелиальных клеток можно использовать ICAM.
Для гемопоэтических клеток можно использовать IFN-бета или CD45.
Для остеобластов можно использовать OG-2.
[536] Промотор, используемый для нацеливания направляющей РНК, может включать:
промотор РНК-полимеразы III, такой как U6 или H1,
использование промотора РНК-полимеразы II и интронных кассет экспрессии направляющей РНК.
Аденоассоциированный вирус (AAV)
[537] Нацеленный на нуклеиновую кислоту эффекторный белок (такой как белок типа V, такой как C2c2) и одна или более направляющих РНК могут быть доставлены с использованием аденоассоциированного вируса (AAV), лентивируса, аденовируса или других плазмидных или вирусных типов векторов, в частности, с использованием лекарственных форм и доз, приведенных, например, в заявках на патент США №№8454972 (лекарственные формы, дозы для аденовируса), 8404658 (лекарственные формы, дозы для AAV) и 5846946 (лекарственные формы, дозы для ДНК-плазмид) и клинических испытаниях и публикациях на тему клинических испытаний, включающих лентивирус, AAV и аденовирус. Для примера, для AAV способ введения, лекарственная форма и доза приведены в заявке на патент США №8454972 и в клинических испытаниях, включающих AAV. Для аденовируса способ введения, лекарственная форма и доза приведены в заявке на патент США №8404658 и в клинических испытаниях, включающих аденовирус. Для доставки плазмиды способ введения, лекарственная форма и доза приведены в заявке на патент США №5846946 и в клинических испытаниях, включающих плазмиды. Дозы могут быть разработаны на основе среднестатистического человека весом 70 кг (например, взрослого мужчину), и могут быть адаптированы для пациентов, индивидов, млекопитающих различного веса и вида. Частота введения находится в пределах компетенции медицинского или ветеринарного практика (например, врача, ветеринара), в зависимости от обычных факторов включая возраст, пол, общее состояние здоровья, другие состояния пациента или индивида, и конкретное представляющее интерес состояние и симптомы. Вирусные векторы могут быть введены в ткань-мишень. Для специфической для типа клетки модификации генома/транскриптома экспрессия нацеленного на нуклеиновую кислоту эффекторного белка (такого как белок типа V, такой как C2c2) может находиться под контролем специфического для типа клетки промотора. Например, специфическая для печени экспрессия может происходить под контролем промотора альбумина, и специфическая для нейронов экспрессия (например, для нацеливания на расстройства центральной нервной системы) может происходить под контролем промотора синапсина I.
[538] С точки зрения доставки in vivo AAV предпочтительнее других вирусных векторов по двум причинам:
- низкая токсичность (возможно, благодаря способу очистки, не требующему ультрацентрифугирования частей клетки, которые могут вызывать иммунную реакцию), и
- низкая вероятность возникновения инсерционного мутагенеза, потому что AAV не интегрируется в геном хозяина.
[539] Предел упаковывания AAV составляет 4,5 или 4,75 т.п.н. Это означает, что нацеленный на нуклеиновую кислоту эффекторный белок (такой как белок типа V, такой как C2c2), а также промотор и терминатор транскрипции должны быть помещены один и тот же вирусный вектор. Следовательно, варианты осуществления изобретения включают использование гомологов нацеленного на нуклеиновую кислоту эффекторного белка (такого как белок типа V, такого как C2c2), которые короче.
[540] Что касается AAV, то AAV может представлять собой AAV1, AAV2, AAV5 или любую комбинацию. Можно выбрать AAV, исходя из клеток, на которые проводят нацеливание; например, можно выбрать серотипы AAV 1, 2, 5 или AAV1, AAV2, AAV5 с гибридным капсидом или любую их комбинацию для нацеливания в клетки головного мозга или нейроны; можно выбрать AAV4 для нацеливания в сердечную ткань, AAV8 пригоден для доставки в печень. В рамках настоящего изобретения предпочтительными являются индивидуальные промоторы и векторы. Соответствие определенных серотипов AAV и типов клеток (см. Grimm, D. et al, J. Virol. 82: 5887-5911 (2008)) является следующим:
Лентивирус
[541] Лентивирусы представляют собой сложные ретровирусы, способные заражать и экспрессировать свои гены как в митотических, так и в постмитотических клетках. Наиболее известным лентивирусом является вирус иммунодефицита человека (ВИЧ), который использует гликопротеины вирусной оболочки других вирусов для нацеливания в широкий диапазон типов клеток.
[542] Лентивирусы могут быть получены следующим образом. После клонирования pCasES10 (который содержит основу трансферной плазмиды лентивируса), HEK293FT при небольшом числе пересевов (p=5) были посеяны в колбе T-75 до 50% смыкания монослоя за день до трансфекции в DMEM с 10% эмбриональной бычьей сывороткой и без антибиотиков. Через 20 часов питательные среды заменяли на среду OptiMEM (без сыворотки) и трансфекция проводили через 4 часа. Клетки трансфицировали 10 мкг лентивирусной плазмиды для переноса (pCasES10) и следующими упаковочными плазмидами: 5 мкг pMD2.G (псевдотип VSV-g) и 7,5 мкг psPAX2 (gag/pol/rev/tat). Трансфекцию проводили в 4 мл OptiMEM со средством доставки катионных липидов (50 мкл Lipofectamine 2000 и 100 мкл реагента Plus). Через 6 часов питательную среду заменяли на DMEM без антибиотика с 10% эмбриональной бычьей сывороткой. В этих способах используется сыворотку во время культивирования, однако предпочтительными являются способы без сыворотки.
[543] Лентивирус может быть очищен следующим образом. Вирусные супернатанты собирали через 48 часов. Супернатанты сначала очищали от дебриса и пропускали через 0,45-мкм фильтр с низким связыванием белков (PVDF). Затем их ультрацентрифугировали в течение 2 часов при скорости 24000 об/мин. Затем осадки с вирусом ресуспендировали в 50 мкл DMEM в течение ночи при 4°C. Потом их были разделяли на аликвоты и сразу же замораживали при -80°C.
[544] В другом варианте осуществления изобретения также предусматривается минимальный вектор на основе лентивируса животного, отличного от примата, основанный на вирусе инфекционной анемии лошадей (EIAV), особенно для генотерапии глаза (см., например, Balagaan, J Gene Med 2006; 8; 275-285). В другом варианте осуществления изобретения также предусматривается RetinoStat®, лентивирусный вектор для генной терапии на основе вируса инфекционной анемии лошадей, экспрессирующий ангиостатические белки эндостатин и ангиостатин, доставляемый через субретинальную инъекцию для лечения возрастной дегенерации желтого пятна (см., например, Binley et al., HUMAN GENE THERAPY 23:980-991 (September 2012)) и этот вектор может быть изменен для использования в систему нацеливания на нуклеиновую кислоту по настоящему изобретению.
[545] В другом варианте осуществления изобретения самоинактивирующиеся лентивирусные векторы с миРНК, нацеленной на экзон, общий для генов tat/rev ВИЧ, локализованной в ядрышке TAR-ловушки и анти-CCR5-специфического рибозима типа "hammerhead" (см, например, DiGiusto et al. (2010) Sci Transl Med 2:36ra43), может использоваться и/ или быть адаптирован для использования в нацеленной на нуклеиновую кислоту системе по настоящему изобретению. Минимум 2,5×106 CD34+ клеток на килограмм веса пациента могут быть получены предварительным стимулированием в течение 16-20 часов в питательной среде X-VIVO 15 (Lonza), содержащей 2 мкМ L-глютамина, фактор стволовых клеток (100 нг/мл), лиганд Flt-3 (Flt-3L) (100 нг/мл) и тромбопоэтин (10 нг/мл) (CellGenix) в плотности 2×106 клеток/мл. Предварительно стимулированные клетки могут быть трансдуцированы лентивирусным вектором при множественности инфекции, равной 5, в течение 16-24 часов в 75-см2 колбах для культивирования тканей, покрытых фибронектином (25 мг/см2) (RetroNectin, Takara Bio Inc.).
[546] Лентивирусые векторы описаны для лечения для болезни Паркинсона, см., например, публикацию патентных заявок США №20120295960, №7303910 и №7351585. Лентивирусые векторы описаны как способ лечения заболеваний глаз, см., например, публикации патентных заявок США №20060281180, 20090007284, US 20110117189; US 20090017543; US 20070054961, US 20100317109. Лентивирусные векторы были также описаны как средство доставки в мозг, см., например, публикации патентных заявок США № US 20110293571; US 20110293571, US 20040013648, US 20070025970, US 20090111106 и патент США № US 7259015.
Доставка РНК
[547] Доставка РНК: нацеленный на нуклеиновую кислоту белок Cas, например, белок типа V, такой как C2c2, и/или направляющая РНК, могут также быть доставлены в форме РНК. мРНК нацеленного на нуклеиновую кислоту белка Cas (например, белка типа V, такого как C2c2) может быть синтезирована с использованием ПЦР-кассеты, содержащей следующие элементы: промотор T7-последовательность Козака (GCCACC)-эффекторный белок-3'-нетранслируемая область (UTR) полиА-последовательности бета-глобина (последовательность из 120 или более остатков аденина). Такая кассета может быть использована для транскрипции с участием Т7-полимеразы. Направляющие РНК могут также быть транскрибированы путем транскрипции in vitro с кассеты, содержащей промотор T7-GG-последовательность направляющей РНК.
[548] Для усиления экспрессии и уменьшения возможной токсичности последовательность, кодирующая нацеленный на нуклеиновую кислоту эффекторный белок, и/или направляющая РНК могут быть модифицированы так, чтобы содержать один или более модифицированных нуклеозидов, например, с использованием псевдо-U или 5-метил-C.
[549] Способы доставки мРНК в настоящее время представляются особенно перспективными для доставки в печень.
[550] Значительные клинические усилия по доставке РНК были сфокусированы на РНК-i или антисмысловых молекулах, однако такие системы могут быть адаптированы для доставки РНК с целью осуществления настоящего изобретения. В соответствии с этим следует прочесть ссылки на РНК-I и т.д., приведенные ниже.
Системы доставки частиц и/или составов:
[551] Несколько типов систем доставки частиц и/или составов нашли применение в широком спектре биомедицинских применений. В целом, частица определяется как малый объект, ведущий себя как целая единица в отношении ее перемещения и свойств. Далее частицы подразделяются в соответствии с диаметром. Крупные частицы соответствуют диапазону от 2500 до 10000 нанометров. Тонкие частицы имеют размер от 100 до 2500 нанометров. Ультратонкие частицы или наночастицы в целом от 1 до 100 нанометров в размере. Основой для выбора предела в 100 нм является проявление новых свойств, отличающих частицы от насыпного материала, которые обычно развиваются при переходном масштабе длины менее 100 нм.
[552] Как используют в настоящем описании, система доставки частиц/лекарственная форма определяется как любая биологическая система доставки/лекарственная форма, которая включает частицу в соответствии с настоящим изобретением. Частица в соответствии с настоящем изобретением представляет собой любой объект, имеющий самое большое измерение (например, диаметр) менее 100 микронов (мкм). В некоторых вариантах осуществления изобретения частицы по изобретению имеют самое большое измерение менее 10 мкм. В некоторых вариантах осуществления частицы по изобретению имеют самое большое измерение меньше, чем 2000 нанометров (нм). В некоторых вариантах осуществления частицы по изобретени имеют самое большое измерение меньше, чем 1000 нанометров (нм). В некоторых вариантах осуществления частицы по изобретению имеют самое большое измерение меньше, чем 900 нм, 800 нм, 700 нм, 600 нм, 500 нм, 400 нм, 300 нм, 200 нм или 100 нм. Как правило, частицы по изобретению имеют самое большое измерение (например, диаметр) 500 нм или меньше. В некоторых вариантах осуществления частицы по изобретению имеют самое большое измерение (например, диаметр) 250 нм или меньше. В некоторых вариантах осуществления частицы по изобретению имеют самое большое измерение (например, диаметр) 200 нм или меньше. В некоторых вариантах осуществления частицы по изобретению имеют самое большое измерение (например, диаметр) 150 нм или меньше. В некоторых вариантах осуществления изобретения разработанные частицы имеют самое большое измерение (например, диаметр) 100 нм или меньше. Меньшие частицы, например, имеющие самое большое измерение 50 нм или меньше используется в некоторых вариантах осуществления изобретения. В некоторых вариантах осуществления изобретения частицы по изобретению имеют самое большое измерение в диапазоне от 25 нм до 200 нм.
[553] Охарактеризацию частиц (включая, например, морфологию, измерение, и т.д.) проводят с использованием ряд различных способов. Общие способы представляют собой электронную микроскопию (ТЕМ, SEM), атомно-силовую микроскопию (AFM), динамическое рассеяние света (DLS), рентгеновскую фотоэлектронную спектроскопию (XPS), порошковую рентгеновскую дифракцию (XRD), инфракрасную спектроскопию с преобразованием Фурье (FTIR), времяпролетную матрично-активированную лазерную десорбцию/ионизацию (MALDI-TOF), оптическую спектроскопию, интерферометрию двойной поляризации и ядерный магнитный резонанс (NMR). Охарактеризация (измерение размеров) может быть проведена относительно нативных частиц (т.е. перед загрузкой) или после загрузки груза (в настоящем описании груз относится, например, к одному или более компонентам системы CRISPR-Cas, например, ферменту CRISPR, или мРНК, или направляющей РНК, или любой их комбинации, и может включать дополнительные носители и/или эксципиенты), чтобы обеспечить частицы оптимального размера для доставки в любом способе применения настоящего изобретения in vitro, ex vivo и/или in vivo. В определенных предпочтительных вариантах осуществления изобретения охарактеризация размеров частиц (например, диаметра) основана на измерении с использованием динамического рассеяния света (DLS). Упоминаются заявка на патент США №8709843; заявка на патент США №6007845, патент США №5855913; патент США №5985309; заявка на патент США №5543158; и публикация James E. Dahlman and Carmen Barnes et al. Nature Nanotechnology (2014) опубликованная через интернет 11 мая 2014 года, doi:10.1038/nnano.2014.84, касающаяся частицы, способов их получения и применения и измерения.
[554] Системы доставки частиц в рамках настоящего изобретения могут быть представлены в любой форме, включая, но не ограничиваясь ими, твердую, полутвердую, эмульсию или коллоидные частицы. По существу любые такие системы доставки, описанные в настоящем описании, включая, но не ограничиваясь ими, например, системы, основанные на липидах, липосомах, мицеллах, микровезикулах, экзосомах или генных пушках, могут быть предоставлены для доставки частиц в рамках настоящего изобретения.
Частицы
[555] мРНК фермента CRISPR и направляющая РНК могут быть доставлены одновременно с использованием частиц или липидных вирусных оболочек; например, фермент CRISPR и РНК по изобретению, например, в виде комплекса, могут быть доставлены с помощью частиц согласно Dahlman et al., WO 2015089419 A2 и документам, цитируемых в них, таких как 7C1 (см., например, работу James E. Dahlman and Carmen Barnes et al. Nature Nanotechnology (2014) опубликованную через интернет 11 мая 2014 года, doi:10.1038/nnano.2014.84), например, частица доставки, включающая липид или липидоид и гидрофильный полимер, например, катионный липид, и гидрофильный полимер, например, такой, где катионный липид включает 1,2-диолеоил-3-триметиламмоний-пропан (DOTAP) или 1,2-дитетрадеканоил-sn-глицеро-3-фосфохолин (DMPC) и/или где гидрофильный полимер включает этиленгликоль или полиэтиленгликоль (ПЭГ); и/или где частица далее включает холестерин (например, частица состава 1=DOTAP 100, DMPC 0, ПЭГ 0, холестерин 0; состава номер 2=DOTAP 90, DMPC 0, ПЭГ 10, холестерин 0; состава номер 3=DOTAP 90, DMPC 0, ПЭГ 5, холестерин 5), где частицы сформированы c использованием эффективного, многоступенчатого процесса, где сначала, эффекторный белок и РНК смешивают вместе, например, в молярном отношении 1:1, например, при комнатной температуре, например, в течение 30 минут, например, в стерильном, свободном от нуклеаз 1X PBS; и отдельно, DOTAP, DMPC, ПЭГ и холестерин, как применимо для состава, растворяют в спирте, например, 100% этаноле; и, эти два раствора смешивают вместе, чтобы сформировать частицы, содержащие комплексы).
[556] мРНК нацеленных на нуклеиновые кислоты эффекторных белков (таких как белки типа VI, таких как C2c2) и направляющая РНК могут быть доставлены одновременно с использованием частиц или липидов вирусных оболочек.
[557] Например, Su X, Fricke J, Kavanagh DG, Irvine DJ ("In vitro and in vivo mRNA delivery using lipid-enveloped pH-responsive polymer nanoparticles" Mol Pharm. 2011 Jun 6; 8(3):774-87. doi: 10.1021/mp100390w. Epub 2011 Apr 1) описывают биоразлагаемые структурированные частицы c ядром из поли(β-аминоэфира) (PBAE), окруженным фосфолипидным бислоем. Они были разработаны для доставки мРНК in vivo. pH-зависимый компонент PBAE был выбран, чтобы способствовать эндосомному разрушению, в то время как поверхностный липидный слой был выбран, чтобы минимизировать токсичность ядра поликатиона. Следовательно, такой способ предпочтителен для доставки РНК по настоящему изобретению.
[558] В одном варианте осуществления изобретения рассмотрены частицы на основе самосборки биоадгезивных полимеров, которые могут быть применены для доставки пептидов в головной мозг различными способами доставки: пероральным, внутривенным, назальным. Также предусматриваются другие варианты осуществления, такие как пероральная и окулярная доставка гидрофобных лекарств. Технология молекулярной оболочки включает спроектированную полимерную оболочку, которая защищена и доставлена к области заболевания (см., например, Mazza, M. et al. ACSNano, 2013. 7(2): 1016-1026; Siew, A., et al. Mol Pharm, 2012. 9(1): 14-28; Lalatsa, A., et al. J Contr Rel, 2012. 161(2): 523-36; Lalatsa, A., et al., Mol Pharm, 2012. 9(6): 1665-80; Lalatsa, A., et al. Mol Pharm, 2012. 9(6): 1764-74, Garrett, N.L., et al. J Biophotonics, 2012. 5(5-6): 458-68; Garrett, N.L., et al. J Raman Spect, 2012. 43(5): 681-688, Ahmad, S., et al. J Royal Soc Interface 2010. 7: S423-33; Uchegbu, I.F. Expert Opin Drug Deliv, 2006. 3(5): 629-40; Qu, X. et al. Biomacromolecules, 2006. 7(12): 3452-9 и Uchegbu, IF., et al. Int J Pharm, 2001. 224:185-199). Предусматриваются дозы приблизительно 5 мг/кг с единичными или множественными дозами, в зависимости от ткани-мишени.
[559] В одном варианте осуществления частицы, которые могут доставить РНК в злокачественную клетку, чтобы остановить рост опухоли, разработанные лабораторией Dan Anderson в MIT, могут использоваться/и или адаптированы для нацеленной на нуклеиновую кислоту системы по настоящему изобретению. В частности, лаборатория Anderson разработала полностью автоматизированные, комбинаторные системы для синтеза, очистки, охарактеризации и составляения новых биоматериалов и нанолекарственных форм. См., например, Alabi et al., Proc Natl Acad Sci USA. 2013 Aug 6; 110(32): 12881-6; Zhang et al., Adv Mater. 2013 Sep 6; 25(33): 4641-5; Jiang et al., Nano Lett. 2013 Mar 13; 13(3): 1059-64; Karagiannis et al., ACS Nano. 2012 Oct 23; 6(10): 8484-7; Whitehead et al., ACS Nano. 2012 Aug 28; 6(8):6922-9 and Lee et al., Nat Nanotechnol. 2012 Jun 3, 7(6): 389-93.
[560] Заявка на патент США 20110293703 касается липидоидных комплексов, особенно полезных при введении полинуклеотидов, которые могут быть применены для доставки нацеленной на нуклеиновую кислоту системы по настоящему изобретению. В одном аспекте аминоспиртовые липидоидные соединения комбинируют с агентом для доставки в клетку или индивидууму для формирования микрочастиц, наночастиц, липосом или мицелл. Агент, доставляемый частицами, липосомами или мицеллами, может быть в форме газа, жидкости или твердого тела, и агент может быть полинуклеотидом, белком, пептидом или низкомолекулярным соединением. Аминоспиртовые липидоидные соединения могут быть объединены с другими аминоспиртовыми липидоидными соединениями, полимерами (синтетическими или естественными), поверхностно-активными веществами, холестерином, углеводами, белками, липидами, и т.д. для формирования частиц. Затем эти частицы могут быть произвольно объединены с фармацевтическим наполнителем для получения фармацевтической композиции.
[561] В заявке на патент США №20110293703 также описаны способы получения аминоспиртовое липидоидное соединение. Одному или более эквивалентам амина позволяют реагировать с одним или более эквивалентами соединений с эпоксидной концевой группой, что при подходящих условиях позволяет сформировать аминоспиртовое липидоидное соединение по настоящему изобретению. В некоторых вариантах осуществления изобретения все аминогруппы амина полностью реагируют с эпоксидными концевыми группами с образованием третичных аминов. В других вариантах осуществления изобретения все аминогруппы амина не полностью реагируют с эпоксидными концевыми группами, таким образом, приводя к образованию первичных или вторичных аминов в аминоспиртовом липидоидном соединении. Эти первичные или вторичные амины сохраняют способность реагировать с другими электрофилами, такими как различные соединения с эпоксидной концевой группой. Как может оценить специалист в данной области, реакция амина с недостатком соединения с эпоксидными группами приводит ко множеству различных аминоспиртовых липидоидных соединений с различным числом хвостовых частей. Определенные амины могут быть полностью функционализированы двумя соединениями с эпоксидной концевой группой с образованием двух хвостовых частей, в то время как другие молекулы не являются полностью функционализированными соединениями с эпоксидной концевой группой с образованием хвостовых частей. Например, диамин или полиамин могут включать один, два, три, или четыре полученных из соединений с эпоксидной концевой группой хвостовых частей, присоединенных к разным аминогруппам молекулы, что приводит к образованию первичных, вторичных, и третичных аминов. В некоторых вариантах осуществления изобретения все аминогруппы не полностью функционализируются. В некоторых вариантах осуществления изобретения используются два соединения с эпоксидной концевой группой одного типа. В других вариантах осуществления изобретения используются два или более различных соединений с эпоксидными концевыми группами. Синтез аминоспиртовых липидоидных соединений может происходить с растворителем или без него, и синтез может быть выполнен при более высоких температурах в пределах 30-100°C, предпочтительно приблизительно при 50-90°C. Подготовленные аминоспиртовые липидоидные соединения необязательно могут быть очищены. Например, смесь аминоспиртовых липидоидных соединений может быть очищена, чтобы обеспечить аминоспиртовое липидоидное соединение с конкретным числом полученных из эпоксида составных хвостовых частей. Или смесь может быть очищена, чтобы привести к определенному стерео- или региоизомеру. Аминоспиртовые липидоидные соединения также могут быть алкилированы с использованием алкилгалогенида (например, метилиодида) или другого алкилирующего агента, и/или они могут быть ацилированы.
[562] В заявке на патент США №20110293703 также описаны библиотеки аминоспиртовых липидоидных соединений, полученных способами по изобретению. Эти аминоспиртовые липидоидные соединения могут быть получены и/или исследованы с использованием высокопроизводительных способов, включающих жидкостные манипуляторы, роботы, микропланшеты для титрования, компьютеры, и т.д. В некоторых вариантах осуществления изобретения аминоспиртовые липидоидные соединения подвергают скринингу в отношении их способности трансфицировать полинуклеотиды или другие агенты (например, белки, пептиды, маленькие молекулы) в клетку.
[563] Заявка на патент США №20130302401 касается класса поли(бета-аминоспиртов) (PBAA), которые получают с помощью комбинаторной полимеризации. Разработанные PBAA могут использоваться в биотехнологии и биомедицинских способах применения как покрытия (такие как покрытия пленок или многослойных пленок для медицинских устройств или имплантатов), добавки, материалы, наполнители, агенты, не подверженные биологическому обрастанию, агенты для микроструктурирования и агенты клеточной инкапсуляции. Использование PBAA в качестве поверхностных покрытий позволило индуцировать разные уровни воспаления, как in vitro, так in vivo, в зависимости от химической структуры. Большое химическое разнообразие этого класса материалов позволило идентифицировать полимерные покрытия, которые ингибируют активацию макрофагов in vitro. Кроме того, эти покрытия уменьшают привлечение клеток воспаления и уменьшают фиброз после подкожной имплантации карбоксилированных микрочастиц полистирола. Эти полимеры могут использоваться для формирования капсулы комплекса полиэлектролита для инкапсуляции клетки. У изобретения может также быть много других биологических способов применения, таких как антибактериальные покрытия, доставка ДНК или миРНК, и тканевая инженерия на основе стволовых клеток. Рекомендации заявки на патент США №20130302401 могут быть применены к нацеленной на нуклеиновые кислоты системе по настоящему изобретению.
[564] В другом варианте осуществления изобретения предусматриваются липидные наночастицы (LNP). Малая интерферирующая РНК против антитранстиретина была заключена в капсулу липидных наночастиц и доставлена в человеческие клетки (например, см. Coelho et al., N Engl J Med 2013; 369:819-29), и такая система может быть а.даптирована и применена для нацеленной на нуклеиновую кислоту системы по настоящему изобретению. Рассмотренные дозы составляют от приблизительно 0,01 до приблизительно 1 мг на кг массы тела при доставке внутривенно. Для снижения риска инфузионных реакций предусматриваются такие лекарственные препараты как дексаметазон, ацетаминофен, дифенгидрамин, цетиризин и ранитидин. Рассмотрено применение в виде множественных доз, составляющих приблизительно 0,3 мг на килограмм массы тела каждые 4 недели до пяти доз.
[565] Показана высокая эффективность LNP для доставки миРНК в печень (см, например, Tabemero et al, Cancer Discovery, April 2013, Vol. 3, No. 4, pages 363-470), следовательно, LNP могут использоваться для доставки РНК, кодирующей нацеленный на нуклеиновую кислоту эффекторный белок к печени. Дозировка может составлять приблизительно четыре дозы по 6 мг/кг LNP каждые две недели. Tabemero et al. продемонстрировали, что регресс опухоли наблюдался после первых 2 циклов LNP при дозировке на уровне 0,7 мг/кг, к концу 6 циклов пациент достиг частичной ремиссии с полной регрессией метастазов лимфатических узлов и существенным уменьшением опухолей печени, полная ремиссия была достигнута после 40 доз, потом ремиссия сохранилась, лечение было закончено после 26 месяцев. У двух пациентов с RCC и внепеченочными участками заболевания, включая почку, легкое и лимфатические узлы, которые прогрессировали после предшествующей терапии с ингибиторами каскада VEGF, было стабильное заболевание во всех областях в течение приблизительно 8-12 месяцев, и пациент с PNET и метастазами печени продолжил лечение в дополнительном исследовании в течение 18 месяцев (36 доз) со стабильным заболеванием.
[566] Однако заряд LNP также должен быть принят во внимание. Катионные липиды комбинируются с отрицательно заряженными липидами для индукции небислойных структур для облегчения внутриклеточной доставки. Поскольку заряженные LNP быстро покидают кровоток после внутривенной инъекции, были разработаны ионизируемые катионные липиды со значениями pKa ниже 7 (см., например, Rosin et al, Molecular Therapy, vol. 19, no. 12, pages 1286-2200, Dec. 2011). Отрицательно заряженные полимеры, такие как РНК могут быть загружены в LNP при низких значениях pH (например, pH 4), когда ионизируемые липиды имеют положительный заряд. Однако при физиологических значениях pH LNP имеют низкий поверхностный заряд, совместимый с более длительным временем циркуляции. Исследование было сосредоточено на четырех видах ионизируемых катионных липидов, а именно, 1,2-дилинеоил-3-диметиламмоний-пропан (DLinDAP), 1,2-дилинолеилокси-3-N, N-диметиламинопропан (DLinDMA), 1,2-дилинолеилокси-кето-N,N-диметил-3-аминопропан (DLinKDMA) и 1,2-дилинолеил-4-(2-диметиламиноэтил)-[1,3]-диоксолан (DLinKC2-DMA). Было показано, что системы миРНК LNP, содержащие эти липиды, показывают значительно отличающиеся свойства подавления экспрессии генов в гепатоцитах in vivo с эффективностью, варьирующей согласно ряду DLinKC2-DMA>DLinKDMA>DLinDMA>>DLinDAP при использовании моделей подавления экспрессии гена фактора VII (см., например, Rosin et al, Molecular Therapy, vol, 19, no. 12, pages 1286-2200, Dec, 2011). Может быть рассмотрена дозировка 1 мкг/мл LNP или РНК CRISP-Cas внутри или в ассоциации с LNP, особенно для лекарственного препарата, содержащего DLinKC2-DMA.
[567] Подготовка LNP и инкапсуляция CRISPR-Cas может быть использована или адаптирована из Rosin et al, Molecular Therapy, vol. 19, no. 12, pages 1286-2200, Dec, 2011. Катионные липиды 1,2-дилинеоил-3-диметиламмоний-пропан (DLinDAP), 1,2-дилинолеилокси-3-N, N-диметиламинопропан (DLinDMA), 1,2-дилинолеилокси-кето-N,N-диметил-3-аминопропан (DLinKDMA), 1,2-дилинолеил-4-(2-диметиламиноэтил)-[1,3]-диоксолан (DLinKC2-DMA), (3-o-[2''-(метоксиполиэтиленгликоль 2000)сукциноил]-1,2-димиристоил-sn-гликоль (ПЭГ-S-DMG) и R-3-[(ко-метокси-поли(этиленгликоль)2000) карбамоил]-1,2-димиристилоксипропил-3-амин (ПЭГ-C-DOMG) могут быть приобретены у Tekmira Pharmaceuticals (Ванкувер, Канада) или синтезированы. Холестерин может быть приобретен у Sigma (Сент-Луис, Миссури). РНк конкретного нацеленного на нуклеиновую кислоту комплекса (CRISPR-Cas) может быть заключена в капсулу из LNP, содержащую DLinDAP, DLinDMA, DLinK-DMA и DLinKC2-DMA (катионный липид:DSPC:CHOL:PEGS-DMG или ПЭГ-C-DOMG в молярных отношениях 40:10:40:10). При необходимости 0,2% SP-DiOC18 (Invitrogen, Берлингтон, Канада) может быть включен в состав, чтобы оценить клеточный захват, внутриклеточную доставку и биораспределение. Инкапсуляция может быть выполнена путем растворения липидной смеси, состоящей из катионного липида:DSPC:холестерола:ПЭГ-c-DOMG (молярное отношение 40:10:40:10), в этаноле до конечной концентрации липида 10 ммоль/л. Этот раствор липида в этаноле может быть добавлен по каплям к цитрату с концентрацией 50 ммоль/л, pH 4,0, для формирования многослойных везикул в целях достижения конечной концентрации этанола 30% по объему. Большие однослойные везикулы могут быть сформированы после пропускания многослойных везикул через два сложенных поликарбонатных фильтра Nuclepore на 80 нм с использованием экструдера (Northern Lipids, Ванкувер, Канада). Инкапсуляция может быть достигнута добавлением РНК, растворенной до концентрации 2 мг/мл, в 50 ммоль/л цитрата, pH 4,0, содержащего 30% этанола по объему, по каплям к предварительно сформированным большим однослойным пузырькам и инкубации при температуре 31°C в течение 30 минут с постоянным перемешиванием до конечного отношения РНК/липид по весу, равного 0,06/1. Удаление этанола и нейтрализация полученного состава буфером могут быть выполнены с помощью диализа против натрий-фосфатного буфера (PBS), pH 7,4, в течение 16 часов с использованием восстановленных целлюлозных мембран для диализа Spectra/Por 2. Гранулометрический состав может быть определен динамическим рассеянием света с использованием гранулометра NICOMP 370, режима везикул/интенсивности и Гауссовской аппроксимации (Nicomp Particle Sizing, Санта-Барбара, Калифорния). Размер частиц для всех трех систем LNP может составлять ~70 нм в диаметре. Эффективность инкапсуляции РНК может быть определена удалением свободной РНК с использованием колонки VivaPureD Mini (Sartorius Stedim Biotech) из образцов, собранных до и после диализа. Инкапсулированная РНК может быть извлечена из элюированных частиц и определена количественно на уровне 260 нм. Отношение РНК к липидам может быть определено измерением содержания холестерина в везикулах с использованием ферментативного анализа с холестерином E от Wako Chemicals USA (Ричмонд, Вирджиния). Вместе с обсуждаемыми в настоящем описании LNP и липидами ПЭГ, пегилированные липосомы или LNP так же подходят для доставки нацеленной на нуклеиновую кислоту системы или ее компонентов.
[568] Получение липидных наночастиц (LNP) возможно производить согласно и/или адаптировано из Rosin et al, Molecular Therapy, vol. 19, no. 12, pages 1286-2200, Dec. 2011. Предварительно подготовленный раствор липидной смеси (с общей концентрацией липидов 20,4 мг/мл) может быть получен в этаноле, содержащем DLinKC2-DMA, DSPC и холестерин в молярных соотношениях 50:10:38,5. Ацетат натрия может быть добавлен в предварительно подготовленный раствор липидной смеси в молярном соотношении 0,75:1 (ацетат натрия: DLinKC2-DMA). Липиды могут быть далее гидратированы с использованием смеси 1,85 объема цитратного буфера (10 ммоль/л, pH 3,0) при активном перемешивании, приводящем к спонтанному образования липосом в водном буфере, содержащем 35% этанол. Раствор липосом может быть инкубирован при 37°C, чтобы сделать возможным зависимое от времени увеличение размеров частиц. Аликвоты могут быть удалены в разные моменты времени в ходе инкубации для исследования изменений размера липосом с помощью способа динамического светорассеяния (Zetasizer Nano ZS, Malvern Instruments, Worcestershire, Великобритания). При достижении желаемого размера частиц водный раствор ПЭГ и липидов (исходный раствор=10 мг/мл ПЭГ-DMG в 35% (объем/объем) этанола) может быть добавлен к смеси липосом для достижения конечной концентрации ПЭГ 3,5% от общего липида. При добавлении ПЭГ-липидов липосомы должны быть примерно их размера и эффективно подавляя дальнейший рост. Далее РНК может быть добавлена к пустым липосомам при соотношении РНК к общим липидам примерно 1:10 (вес:вес) с последующей инкубацией на протяжении примерно 30 мин при 37°C для образования загруженных липидных наночастиц (LNP). Такая смесь может быть в дальнейшем подвергнута диализу на протяжении ночи в PBS) и фильтрации через 0,45-пм фильтр-шприц.
[569] Конструкции сферических нуклеиновых кислот (Spherical Nucleic Acid, SNA™) и другие частицы (в частности, частицы золота) также предусматриваются в качестве средства доставки нацеленных на нуклеиновую кислоту систем к предполагаемым мишеням. Значительный объем данных демонстрирует полезность конструкций терапевтических сферических нуклеиновых кислот от AuraSense (SNA™), основанных на функционализированных нуклеиновой кислотой золотых частицах.
[570] Литература, которая может быть использована в совокупности с изложенными в настоящем описании идеями, включает: Cutler et al., J. Am. (Chem. Soc. 2011 133:9254-9257, Hao et al., Small. 2011 7:3158-3162, Zhang et al., ACS Nano. 2011 5:6962-6970, Cutler et al., J. Am. Chem. Soc. 2012 134:1376-1391, Young et al., Nano Lett. 2012 12:3867-71, Zheng et al., Proc. Natl. Acad. Sci. USA. 2012 109:11975-80, Mirkin, Nanomedicine 2012 7:635-638 Zhang et al., J. Am. Chem. Soc. 2012 134:16488-1691, Weintraub, Nature 2013 495:S14-S16, Choi et al., Proc. Natl. Acad. Sci. USA. 2013 110(19):7625-7630, Jensen et al., Sci. Transl. Med. 5, 209ral52 (2013) и Mirkin, et al., Small, 10:186-192.
[571] Самособирающиеся частицы с РНК могут быть произведены из полиэтиленимина (PEI), связанного с ПЭГ через пептидный лиганд Arg-Gly-Asp (RGB), присоединенный к дальнему концу ПЭГ. Такая система была использована, например, в качестве средства для нацеливания на сосуды опухоли, экспрессирующей интегрины, и доставки миРНК, ингибирующей экспрессию рецептора факторов роста эндотелия сосудов 2 (VEGFR2), и достижения, таким образом, ангиогенеза в опухоли (см., например, Sehiffelers et al, Nucleic Acids Research, 2004, Vol. 32, No. 19). Наноплексы могут быть приготовлены смешиванием равных объемов водных растворов катионного полимера и нуклеиновой кислоты для получения чистого молярного избытка ионизируемого азота (полимер) относительно фосфата (нуклеиновая кислота) в диапазоне от 2 до 6. Электростатические взаимодействия между катионными полимерами и нуклеиновой кислотой приводят к образованию полиплексов со средним значением распределения размеров примерно 100 нм, в связи с чем в настоящем описании они названы наноплексами. Предусматривается дозировка примерно от 100 до 200 мг РНК комплекса, нацеленного на нуклеиновую кислоту, для доставки в самособирающихся частицах согласно Sehiffelers et al.
[572] Наноплексы, описанные Bartlett et al. (PNAS, September 25, 2007, vol. 104, no. 39) также могут быть применены в рамках настоящего изобретения. Наноплексы, описанные Bartlett et al., получают смешиванием равных объемов водных растворов катионного полимера и нуклеиновой кислоты для получения чистого молярного избытка ионизируемого азота (полимера) по отношению к фосфату (нуклеиновая кислота) в диапазоне от 2 до 6. Электростатические взаимодействия между катионными полимерами и нуклеиновой кислотой приводят к образованию полиплексов со средним значением распределения размеров примерно 100 нм, в связи с чем здесь они названы наноплексами. DOTA-миРНК, описанная Bartlett et al., была синтезирована следующим образом: 1,4,7,10-тетразациклододекан-1,4,7,10-тетрауксусной кислоты моно (N-гидроксисукцинимид эфир) (DOTA-эфир NHS) был заказан у Macrocyclics (Даллас, Техас, США). Кодирующая цепь аминомодифицированной РНК со 100-кратным молярным избытком эфира DOTA-NHS в карбонатном буфере (pH 9) была помещена в пробирку для микроцентрифугирования. Реакция содержимого была проведена при перемешивании на протяжении 4 ч при комнатной температуре. Связанные DOTA-кодирующая РНК были осаждены этанолом, ресуспенидрованы в воде и прошли отжиг на немодифицированную некодирующую цепь для получения DOTA-миРНК. Все жидкости были предварительно обработаны Chelex-100 (Bio-Rad, Hercules, Калифорния, США) для удаления следов загрязнения металлом. Нацеленные на Tf и ненацеленные частицы миРНК могут быть образованы с использованием содержащих циклодекстрин поликатионов. Обычно частицы образуются в воде при соотношении зарядов (+/-), равном 3, и концентрации миРНК 0,5 г/л. Один процент молекул адамантан-ПЭГ на поверхности нацеленных частиц был модифицирован (адамантан-ПЭГ-Tf). Частицы были суспендированы в 5% (масса/объем) полученного раствора переносчика глюкозы для инъекций.
[573] Davis et al. (Nature, Vol 464, 15 April 2010) провели клинические испытания РНК с использованием системы доставки нацеленных частиц (регистрационный номер клинического испытания NCT00689065). Пациенты с солидным типом злокачественной опухли, нечувствительной к стандартным способам лечения, получали дозировки направленных частиц в дни 1, 3, 8 и 10 21-дневного курса путем 30-минутного внутривенного вливания. Такие частицы включают, по существу состоят или состоят из синтетической системы доставки, содержащей: (1) линейный полимер на основе циклодекстрина (CDP), (2) лиганд, нацеленный на белок трансферрин человека (TF), находящийся снаружи наночастицы для активации рецепторов TF (TFR) на поверхности злокачественных клеток, (3) гидрофильный полимер (полиэтиленгликоль, ПЭГ) используется для увеличения стабильности наночастиц в биологических жидкостях) и (4) миРНК, разработанная для уменьшения экспрессии RRM2 (используемая в клинической практике последовательность ранее была обозначена как siR2B+5). На протяжении долгого времени было известно, что TFR активнее экспрессируется в клетках злокачественных опухолей, поэтому RRM2 является доказанная мишень терапии против злокачественной опухоли. Такие частицы (используемая в клинической практики версия обозначена как CALAA-01) продемонстрировали хорошую переносимость в исследованиях с различными дозировками на отличных от человека приматах. Хотя ранее один пациент с хроническим миелоидным лейкозом получал миРНК путем доставки липосомами, клинические испытания Davis et al. являются первичными для систематической доставки миРНК с нацеленной системой доставки и с целью лечения пациентов с солидными злокачественными опухолями. Для проверки того, может ли система направленной доставки обеспечить эффективную доставку функциональной миРНК в опухоли у человека, Davis et al. исследовали материалы биопсии трех пациентов из трех групп по дозировке: пациенты A, В и C, каждый из которых имел метастазирующую меланому и получал CALAA-01 в дозах 18, 24 и 30 мг/м2 миРНК соответственно. Сходные дозы могут также быть предположены для нацеленной на нуклеиновую кислоту системы по настоящему изобретению. Доставка согласно изобретению может быть достигнута с использованием частиц, содержащих линейный полимер на основе циклодекстрина (CDP), лиганд, нацеленный на белок трансферрин человека (TF), находящийся снаружи наночастицы для активации рецепторов TF (TFR) на поверхности злокачественных клеток и/или гидрофильный полимер (например, полиэтиленгликоль (ПЭГ), используемый для повышения стабильности в биологических жидкостях).
[574] В отношении настоящего изобретения, предпочтительно наличие одного или более компонентов нацеленного на нуклеиновую кислоту комплекса, например, нацеленного на нуклеиновую кислоту эффекторного белка или мРНК, либо направляющая РНК, доставленных при помощи частиц или липидной упаковки. Другие системы доставки или векторы могут быть использованы в совокупности с частицами в соответствующих вариантах изобретения.
[575] В целом под "наночастицей" понимают любую частицу с диаметром менее 1000 нм. В определенных предпочитаемых вариантах осуществления наночастицы по настоящему изобретению имеют размер вдоль длинной оси (например, в диаметре) 500 нм или менее. В других предпочитаемых вариантах, описываемых в данном изобретении, наночастицы имеют наибольший размер в диапазоне между 25 нм и 200 нм. В других предпочитаемых вариантах описываемые в данном изобретении наночастицы имеют вдоль длинной оси размер 100 нм или менее. В других предпочитаемых вариантах описываемые в данном изобретении наночастицы имеют размер от 35 нм до 60 нм.
[576] Частицы, относящиеся к настоящему изобретению, могут быть доставлены в различных формах, например, твердые частицы (например, металлы как серебро, золото, железо, титан), неметаллы, твердые частицы на основе липидов, полимеры, суспензии частиц или их комбинации. Могут быть получены частицы из металлов, диэлектриков или полупроводников, как и гибридные структуры (например, с ядром и оболочкой). Частицы из полупроводниковых материалов могут быть также маркированы квантовыми точками, если имеют размер, достаточно малый (обычно менее 10 нм) для возможности квантования электронных уровней. Такие наночастицы используются в биомедицинских применениях в качестве носителей для лекарств или агентов для визуализации и могут быть адаптированы для сходных целей в рамках настоящего изобретения.
[577] Были произведены полутвердые и мягкие частицы, входящие в объем настоящего изобретения. Частицей-прототипом полутвердой природы является липосома. Различные типы липосомных частиц в настоящее время используются в клинической практике в качестве систем доставки для противораковых лекарств и вакцин. Наполовину гидрофильные и наполовину гидрофобные частицы называются янус-частицами и они особенно эффективны для стабилизации эмульсий. Они способны к самостоятельной сборке на поверхностях раздела вода/масло и действуют как твердые поверхностно-активные вещества.
[578] В патенте США №8709843, включенном в настоящее описание в качестве ссылки, предложена система доставки лекарств для нацеленной доставки содержащих терапевтический агент частиц к тканям, клеткам и внутриклеточным компартментам. Настоящее изобретение относится к нацеленным частицам, содержащим полимер, связанный с поверхностно-активным веществом, гидрофильный полимер или липид.
[579] В патенте США №6007845, включенном в настоящее описание в качестве ссылки, предложены частицы, имеющие ядро из мультиблочного сополимера, образованное путем ковалентного связывания мультифункционального соединения с одним или более гидрофильными полимерами, и содержащие биологически активный материал.
[580] В патенте США №5855913, включенном в настоящее описание в качестве ссылки, предложена композиция частиц, включающая аэродинамически легкие частицы с плотностью менее 0,4 г/см3 и средним диаметром от 5 пм до 30 пм, имеющих на поверхности поверхностно-активное вещество для доставки лекарств в систему легких.
[581] В патенте США №5985309, включенном в настоящее описание в качестве ссылки, предложены частицы, содержащие поверхностно-активное вещество и/или гидрофильный или гидрофобный комплекс с положительно или отрицательно заряженным лекарственным веществом или агентом для диагностики и заряженную частицу с противоположным зарядом для доставки в легкие.
[582] В патенте США №5543158, включенном в настоящее описание в качестве ссылки, предложены биоразлагаемые инъецируемые частицы с биоразлагаемым твердым ядром, содержащим биологически активный материал и остатки поли(алкиленгликоля) на поверхности.
[583] В WO 2012135025 (также опубликованной как US 20120251560), включенной в настоящем описании в качестве ссылки, описаны полимеры конъюгированного полиэтиленимина (PEI) и конъюгированных азамакроциклов (вместе называемых "конъюгированный липомер" или "липомеры"). В некоторых вариантах осуществления может быть предусмотрено, что такие способы и материалы из цитируемых в настоящем описании документов, например, конъюгированные липомеры, могут быть использованы в контексте нацеленной на нуклеиновую кислоту системы для достижения in vitro, ex vivo и in vivo изменений генома для модификации экспрессии генов, а также экспрессии белков.
[584] В одном варианте осуществления частица может быть модифицированным эпоксидом липидным полимером, предпочтительно 7C1 (см., например, James E. Dahlman and Carmen Barnes et al. Nature Nanotechnology (2014) опубликованная через интернет 11 мая 2014 года, doi:10.1038/nnano.2014.84). C71 был синтезирован в реакции C15 липидов с концевыми эпоксидными группами с PEI600 при молекулярном соотношении 14:1 и составлен с C14PEG2000 для получения частиц (диаметр между 35 и 60 нм), стабильных в растворе PBS (фосфатно-солевой буфер, PBS) на протяжении не менее 40 дней.
[585] Модифицированный эпоксидными группами липид-полимер может быть использован для доставки нацеленной на нуклеиновую кислоту системы по настоящему изобретению в клетки легких, сердечно-сосудистой системы или почек, однако, квалифицированный специалист в данной области может адаптировать систему для доставки к другим органам-мишеням. Предполагаются дозировки в диапазоне от приблизительно 0,05 до приблизительно 0,6 мг/кг. Также предусматриваются дозировки, вводимые на протяжении нескольких суток или недель с общей дозой приблизительно 2 мг/кг.
Экзосомы
[586] Экзосомы являются эндогенными нановезикулами, транспортирующими РНК или белки, которые могут доставлять РНК в головной мозг и другие органы-мишени. Для уменьшения иммуногенности Alvarez-Erviti et al. (2011, Nat Biotechnol 29: 341) использовали полученные ими собственные дендритные клетки мышей для получения экзосом. Нацеливание в головной мозг было достигнуто инженерией дендритных клеток, приводящей к экспрессии Lamp2b, экзосомального мембранного белка, слитого со специфическим пептидом нейронов RVG. Очищенные экзосомы были загружены экзогенной РКН с помощью электропорации. миРНК GAPDH, доставленная введенными внутривенно нацеленными на RVG экзосомами в нейроны, микроглию, олигодендроциты в головном мозге вызывает нокдаун конкретного гена. Предварительная обработка экзосомами RVG не ослабляла нокдаун, неспецифический захват в других тканях не наблюдался. Терапевтический потенциал опосредованной экзосомами доставки миРНК был продемонстрирован сильным нокдауном мРНК (60%) и белка (62%) BACE1, терапевтической мишени болезни Альцгеймера.
[587] Для получения совокупности иммунологически инертных экзосом Alvarez-Erviti et al. взяли пробы костного мозга мышей инбредной линии C57BL/6 с однородным гаплотипом по главному комплексу гистосовместимости (MHC). Поскольку незрелые дендритные клетки производят большие объемы экзосом, лишенных активаторов Т-клеток, таких как MHC-II и CD86, Alvarez-Erviti et al. отбирали дендритные клетки с использованием гранулоцитарно-макрофагального колониестимулирующего фактора (GM-CSF) на протяжении 7 дней. Экзосомы были очищены от культурального супернатанта на следующий день с использованием стандартных протоколов ультрацентрифугирования. Полученные экзосомы были физически однородны с распределением размеров, имеющим пик, соответствующий диаметру 80 нм, как установлено путем анализа траекторий частиц (NTA) и электронной микроскопией. Alvarez-Erviti et al. получили 6-12 мкг экзосом (измерено на основе концентрации белка) на 106 клеток.
[588] Далее Alvarez-Erviti et al. исследовали возможность загрузки модифицированных экзосом экзогенным грузом при помощи протоколов электропорации, адаптированных для применения в наномасштабе. Поскольку электропорация мембранных частиц в наноразмерном масштабе недостаточно охарактеризована, неспецифическая маркированная Cy5 РНК была использована для оптимизации протокола электропорации эмпирически. Количество упакованной РНК было оценено после ультрацентрифугирования и лизиса экзосом. Электропорация при 400 В и 125 пФ привела к максимальному удержанию РНК и использовалась во всех последующих экспериментах.
[589] Alvarez-Erviti et al. вводили 150 мкг каждой миРНК BACE1, упакованной в 150 пг экзосом RVG, нормальным мышам C57BL/6 и сравнивали эффективность нокдауна с четырьмя контролями: интактные мыши, мыши, которым вводились только экзосомы RVG, мыши, которым вводились миРНК BACE1 в комплексе с in vivo реагентом катионных липосом и мыши, которым вводились миРНК BACE1 в комплексе с RVG-9R, пептидом RVG, связанным с 9 остатками D-аргинина, электростатически связанными с миРНК. Образцы кортикальной ткани были проанализированы после 3 дней введения, был обнаружен существенный нокдаун белка (45%, P<0,05, против 62%, P<0,01) у получавших экзосомы как с миРНК-RVG-9R, так и с миРНК-RVG, что было следствием существенного уменьшения уровней мРНК BACE1 (66% [+ или -] 15%, P<0,001 и 61% [+ или -] 13% соответственно, P<0,01). Более того, заявители продемонстрировали существенное уменьшение (55%, P<0,05) общих уровней бета-амилоида 1-42, главного компонента амилоидных бляшек при патологии болезни Альцгеймера в случае животных, которым вводили экзосомы RVG. Наблюдаемое уменьшение превосходило уменьшение для бета-амилоида 1-40 у нормальных мышей после внутрижелудочкового введения ингибиторов BACE1. Alvarez-Erviti et al. осуществили быструю амплификацию 5'-концов кДНК (RACE) для продукта расщепления BACE1, что предоставило доказательства опосредованного РНК-интерференцией нокдауна за счет миРНК.
[590] Наконец, Alvarez-Erviti et al. исследовали, вызывают ли экзосомы РНК-RVG иммунный ответ in vivo путем измерения концентраций L-6, IP-10, TNFα и IFN-α в сыворотке. После введения экзосомам для всех цитокинов были зарегистрированы несущественные изменения, сходные с возникающими при введении реагента для трансфекции миРНК, и противоположные возникающим при обработке миРНК-RVG-9R, которая стимулирует высокую секрецию IL-6, что подтверждает иммунологическую инертность при введении экзосом. Учитывая, что экзосомы содержат только 20% миРНК, доставка RVG-экзосомами представляется более эффективной, чем доставка RVG-9R при достижении сравнимого уровня нокдауна мРНК с использованием меньшего в 5 раз количества миРНК без соответствующего уровня иммунной стимуляции. Данный эксперимент продемонстрировал терапевтический потенциал технологии RVG-экзосом, которая может подходить для долговременного сайленсинга генов, связанных с нейродегенеративными заболеваниями. Система доставки с использованием экзосом по Alvarez-Erviti et al. может быть применена для доставки нацеленной на нуклеиновую кислоту системы по настоящему изобретению к мишеням терапии, особенно в случае нейродегенеративных заболеваний. Доза в диапазоне примерно от 100 до 1000 мг нацеленной на нуклеиновую кислоту системы, упакованной в от ~100 до ~1000 мг RVG-экзосом, может быть предусмотрена в рамках настоящего изобретения.
[591] El-Andaloussi et al. (Nature Protocols 7, 2112-2126(2012)) установили, каким образом полученные из культивируемых клеток экзосомы могут быть использованы для доставки РНК in vitro и in vivo. Данный протокол вначале описывает получение нацеленных экзосом путем трансфекции экспрессирующего вектора, содержащего экзосомальный белок, слитый с пептидным лигандом. Далее El-Andaloussi et al. описывают очистку и охарактеризацию экзосом из супернатанта трансфицированных клеток. Далее El-Andaloussi et al. детализируют ключевые шаги загрузки РНК в экзосомы. В конце El-Andaloussi et al. кратко описывают использование экзосом для эффективной in vitro и in vivo доставки РНК в головной мозг мыши. Примеры ожидаемых результатов, для которых количественно оценена опосредованная экзосомами доставка путем функционального анализа и визуализации, также приведены. Полный протокол занимает ~3 недели. Доставка или введение согласно изобретению могут быть выполнены с использованием экзосом, полученных из дендритных клеток пациента. В соответствии с описанными в настоящем описании идеями, это может быть использовано в практике использования изобретения.
[592] В другом варианте осуществления рассматриваются экзосомы плазмы из Wahlgren et al. (Nucleic Acids Research, 2012, Vol. 40, No. 17 el30). Экзосомы являются наноразмерными везикулами (размером 30-90 нм), производимыми многими типами клеток, включая дендритные клетки (DC), В-клетки, T-клетки, тучные клетки, эпителиальные клетки и клетки опухоли. Такие везикулы образуются путем впячивания и отделения поздних эндосом и далее освобождаются во внеклеточную среду при слиянии с плазматической мембраной. Поскольку экзосомы в естественном состоянии переносят РНК между клетками, такое их свойство может быть полезно для генной терапии, и исходя из данного описания экзосомы могут быть использованы при применении настоящего изобретения.
[593] Экзосомы из плазмы могут быть получены путем центрифугирования фракции тромбоцитов и белых клеток крови при 900g на протяжении 20 мин для выделения плазмы с последующим отбором супернатанта, центрифугирования при 300g на протяжении 10 мин для удаления клеток и при 16500g на протяжении 30 мин с последующей фильтрацией через 0,22-мм фильтр. Экзосомы осаждаются при ультрацентрифугировании при 120000g на протяжении 70 мин. Химическая трансфекция миРНК в экзосомы выполнена согласно рекомендациям производителя для начального набора для РНК-i человек/мышь (RNAi Human/Mouse Starter Kit, Quiagen, Хильден, Германия). миРНК добавляется к 100 мл PBS в конечной концентрации 2 ммоль/мл. После добавления реагента для трансфекции HiPerFect смесь инкубируется на протяжении 10 мин при нормальных условиях. Для того, чтобы удалить избыток мицелл, экзосомы вновь выделяют с использованием альдегид/сульфатных латексных шариков. Химическая трансфекция нацеленной на нуклеиновую кислоту системы в экзосомы может быть проведена сходным образом с таковой для миРНК. Такие экзосомы могут быть сокультивированы с моноцитами и лимфоцитами, выделенными из периферической крови здоровых доноров. Следовательно, можно предположить, что экзосомы, содержащие нацеленную на нуклеиновые кислоты систему, могут быть введены в моноциты и лимфоциты и аутологично вновь введены в человека. В соответствии с этим, доставка или введение в соответствии с изобретением могут быть выполнены с применением экзосом плазмы.
Липосомы
[594] Доставка или введение по изобретению могут быть выполнены с помощью липосом. Липосомы представляют собой сферические везикулярные структуры, состоящие из одно- или многослойного липидного бислоя, окружающего внутренние водные компартменты, и относительно непроницаемого внешнего липофильного фосфолипидного бислоя. Липосомы привлекают значительное внимание как переносчики для доставки лекарственных средств, потому что они биологически совместимы, нетоксичны, могут доставлять и гидрофильные и липофильные молекулы препарата, защитить свой груз от деградации ферментами плазмы и переносить свой груз через биологические мембраны и гемато-энцефалический барьер (ГЭБ) (см., например, Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi: 10.1155/2011/469679 для обзора).
[595] Липосомы могут состоять из нескольких различных типов липидов; однако, для производства липосом в качестве переносчиков препаратов обычно используются фосфолипиды. Липосомы образуются самопроизвольно при смешивании липидной пленки с водным раствором, но этот процесс может быть ускорен с помощью механического воздействия в виде встряхивания при помощи гомогенизатора, генератора ультразвука или экструдера (см.. например, Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi: 10.1155/2011/469679 для обзора).
[596] Несколько других добавок могут быть добавлены к липосомам, чтобы изменить их структуру и свойства. Например, холестерин или сфингомиелин могут быть добавлены к липосомальной смеси для стабилизации структуры липосом и предотвращения потери внутреннего груза. Далее, липосомы могут быть получены из гидрированного фосфатидилхолина яйца или фосфатидилхолина яйца, холестерина и диацетилфосфата, и средние размеры везикул могут быть доведены до приблизительно 50 и 100 нм. (см., например, Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi: 10.1155/2011/469679 для подробного обзора).
[597] Лекарственная форма липосомы может состоять, главным образом, из естественных фосфолипидов и липидов, таких как 1,2-дистеароил-sn-глицеро-3-фосфатидилхолин (DSPC), сфингомиелин, фосфатидилхолины яйца и монозиалоганглиозид. Так как эта лекарственная форма составлена только из фосфолипидов, при создании липосом возникло много проблем, в том числе их нестабильность в плазме. Было предпринято несколько попыток преодолеть эти проблемы, в основном путем изменения состава липидной мембраны. Одна из этих попыток заключалась в добавлении холестерина. Добавление холестерина к обычным лекарственным формам замедляет высвобождение инкапсулированного биологически активного вещества в плазму, или 1,2-диолеоил-sn-глицеро-3-фосфоэтаноламин (DOPE) увеличивает стабильность (см., например, Spuch and Navarro, Journal of Drug Delivery, vol. 2011, Article ID 469679, 12 pages, 2011. doi: 10.1155/2011/469679 для обзора).
[598] В особенно предпочтительном варианте осуществления изобретения желательно использование липосом, действующих по принципу "троянского коня" (также известный как "молекулярные троянские кони"), и протоколы можно найти по ссылке http://cshprotocols.cshlp.org/content/2010/4/pdb.prot5407.long.
Эти частицы обеспечивают доставку трансгена ко всему головному мозгу после внутрисосудистой инъекции. Без ограничения, считается, что нейтральные липидные частицы со специфичными антителами, связанными с их поверхностью, способны пересекать гематоэнцефалический барьера путем эндоцитоза. Заявитель утверждает, что липосомы, действующие по принципу "троянского коня", могут быть использованы для доставки семейства нуклеаз CRISPR к головному мозгу путем внутрисосудистой инъекции, что позволило бы создавать животных с трансгенным головным мозгом без потребности в манипуляции с эмбрионами. Приблизительно 1-5 г ДНК или РНК могут быть использованы для введения in vivo в липосомах.
[599] В другом варианте осуществления изобретения нацеленную на нуклеиновую кислоту систему или ее компоненты можно доставить при помощи липосом, например, стабильной нуклео-липидной частицы (SNALP) (см., например, Morissey et al., Nature Biotechnology, Vol. 23, No. 8, August 2005). Предусматриваются ежедневные внутривенные инъекции приблизительно 1, 3 или 5 мг/кг/сутки конкретной нацеленной на нуклеиновую кислоту системы, нацеленной в SNALP. Ежедневное лечение может быть проведено приблизительно за три дня и затем еженедельно в течение приблизительно пяти недель. В другом варианте осуществления изобретения также предусматривается конкретная нацеленная на нуклеиновую кислоту система, заключенная в SNALP, введенная внутривенной инъекцией в дозах приблизительно 1 или 2,5 мг/кг (см., например, Zimmerman et al., Nature Letters, Vol. 441, 4 May 2006). Состав SNALP может содержать липиды 3-N-[(вметоксиполи(этиленгликоль)2000)карбамоил]-1,2-димиристилокси-пропиламин (ПЭГ-C-DMA), 1,2-дилинолеилокси-N,N-диметил-3-аминопропан (DLinDMA), 1,2-дистеароил-sn-глицеро-3-фосфохолин (DSPC) и холестерин, в соотношении 2:40:10:48 молярных процентов (см., например, Zimmerman et al., Nature Letters, Vol. 441, 4 May 2006).
[600] В другом варианте осуществления было доказано, что стабильные нуклео-липидные частицы (SNALP) являются эффективными молекулами для доставки в сильно васкуляризированные опухоли печени, полученные из HepG2, но не в слабо васкуляризированные опухоли печени, образованные из НСТ-116 (см., например, Li, Gene Therapy (2012) 19, 775-780). Липосомы SNALP могут быть получены смешением D-Lin-DMA и ПЭГ-C-DMA с дистероилфосфатидилхолином (DSPC), холестерином и миРНК, использованных в соотношении 25:1 липид/миРНК и молярным соотношением 48/40/10/2 для холестерина/D-Lin-DMA/DSPC/ПЭГ-C-DMA. Полученные липосомы SNALP имеют размер примерно 80-100 нм.
[601] В еще одном варианте осуществления стабильные частицы нуклеиновая кислота-липид (SNALP) могут включать синтетический холестерин (Sigma-Aldrich, Сент-Луис, Миссури, США), дипальмитоилфосфатидилхолин (Avanti Polar Lipids, Alabaster, Алабама, США), 3-N-[(w-метокси-поли(этиленгликоль)2000)карбамоил]-1,2-димиристоилоксипропиламин и катионный 1,2-дилиеноилокси-3-N,N-диметиламинопропанан (см., например, Geisbert et al., Lancet 2010; 375: 1896-905). Может предусматриваться дозировка примерно 2 мг/кг нацеленной на нуклеиновую кислоту системы, введенная, например, внутривенной болюсной инфузий.
[602] В еще одном варианте осуществления изобретения SNALP может включать синтетический холестерин (Sigma-Aldrich), 1,2-дистеароил-sn-глицеро-3-фосфохолин (DSPC; Avanti Polar Lipids Inc.), ПЭГ-cDMA, и 1,2-дилинолеилокси-3-(N;N-диметил)аминопропан (DLinDMA) (см, например, Judge, J. Clin. Invest. 119:661-673 (2009)). Составы, используемые для исследований in vivo, могут включать конечное соотношение липид/РНК по массе приблизительно 9:1.
[603] Безопасность нанолекарств на основе РНК-i была рассмотрена Barros and Gollob из Alnylam Pharmaceuticals (см., например, Advanced Drug Delivery Reviews 64 (2012) 1730-1737). Стабильная нуклео-липидная частица кислоты (SNALP) состоит из четырех различных липидов: ионизируемого липида (DLinDMA), который является катионным при низких значениях pH, нейтрального липида-помощника, холестерина и способного к диффузии липида на основе полиэтиленгликоля (ПЭГ). Частица составляет приблизительно 80 нм в диаметре и имеет нейтральный заряд при физиологическом pH. Ионизируемый липид служит для связывания липида с анионной РНК во время формирования частицы. При увеличении кислотности внутри эндосомы, ионизируемый липид становится положительно заряженным и опосредует слияние SNALP с мембраной эндосомы, высвобождая РНК в цитоплазму. ПЭГ-содержащий липид стабилизирует частицу и уменьшает агрегирование во время сборки, впоследствии обеспечивая нейтральную гидрофильную поверхность, которая улучшает фармакокинетические свойства.
[604] На данный момент две клинические программы были начаты с использованием смесей SNALP с РНК. Tekmira Pharmaceuticals недавно закончили фазу I клинического испытания с применением единственной дозы SNALP-ApoB на взрослых добровольцах с повышенным уровнем холестерина LDL. ApoB преимущественно экспрессируется в печени и тощей кишке и необходим для сборки и секреции VLDL и LDL. Семнадцать участников исследования получили единственную дозу SNALP-ApoB (постепенное увеличение дозы на протяжении 7 уровней). Не было никаких доказательств токсичности для печени (ожидались как потенциальная ограничивающая дозу токсичность на основе доклинических исследований). Один (из двух) испытуемых при получении самой высокой дозы испытал гриппоподобные симптомы, согласующиеся со стимуляцией иммунной системы, и было принято решение завершить испытание.
[605] Alnylam Pharmaceutcals так же занимались продвижением ALN-TTR01, который использует технологию SNALP, описанную выше, и нацелен на продукцию гепатоцитами мутанта и TTR дикого типа для лечения амилоидоза TTR (ATTR). Были описаны три синдрома ATTR: семейная амилоидная полинейропатия (FAP) и семейная амилоидная кардиомиопатия (FAC) - оба вызываемые аутосомными доминирующими мутациями в TTR; и старческий системный амилоидоз (SSA), вызываемый TTR дикого типа. Недавно была закончена фаза I плацебо-контролируемых испытаний на пациентах с ATTR единственный дозы ALN-TTR01 c ее постепенным увеличением. ALN-TTR01 был введен в виде 15-минутного внутривенного вливания 31 пациенту (23 с препаратом исследования и 8 с плацебо) в диапазоне дозы от 0,01 до 1,0 мг/кг (на основе миРНК). Испытуемые хорошо переносили лечение без значительных увеличений функции печени в тестах. Инфузионные реакции были отмечены в 3 из 23 пациентов при дозе более 0,4 мг/кг; все реагировали на замедление скорости вливания и все продолжили участие в исследовании. Минимальное и временное повышение уровня цитокинов IL-6, IP-10 и IL-1ra в сыворотке крови было отмечено у двух пациентов при самой высокой дозе 1 мг/кг (как предполагалось исходя из доклинических испытаний и исследований на приматах). Понижая уровень TTR в сыворотке, ожидаемый фармакодинамический эффект от введения ALN-TTR01, наблюдался при дозе 1 мг/кг.
[606] В еще одном варианте осуществления изобретения SNALP может быть собран с помощью растворения катионного липида, DSPC, холестерина и ПЭГ-липида, например, в этаноле, например, в молярном отношении 40:10:40:10, соответственно (см., например, Semple et al., Nature Biotechnology, Volume 28 Number 2 February 2010, pp. 172-177). Липидная смесь была добавлена к водному буферу (50-мМ цитрата, pH 4) до достижения окончательной концентрации этанола и липидов 30% (по объему) и 6,1 мг/мл, соответственно, и была уравновешена при 22°C в течение 2 минут перед экструзией. Гидратированные липиды были экструдированы через два сложенных фильтра с размером пор 80 нм (Nuclepore) при 22°C с использованием Lipex Extruder (Northern Lipids) до достижения диаметра везикулы 70-90 нм, как определено динамическим анализом рассеяния света. Это обычно требовало 1-3 проходов. миРНК (растворенная в 50 мМ цитрата, водном растворе с pH 4, содержащем 30% этанола), была добавлена к предварительно уравновешенным (35°C) везикулам со скоростью 5 мл/минуту при смешивании. После достижения окончательного целевого отношения миРНК/липид, равного 0,06 (вес/вес), смесь была инкубирована в течение 30 минут при 35°C, чтобы обеспечить реорганизацию везикул и инкапсуляцию миРНК. Потом этанол был удален и внешний буфер заменен на PBS (155 мМ NaCl, 3 мМ Na2HP04, 1 мМ KH2PO4, pH 7,5) либо диализом, либо фильтрованием из тангенциального потока. миРНК была заключена в капсулу из SNALP с использованием контролируемого пошагового процесса растворения. Липидными компонентами KC2-SNALP были DLin-KC2-DMA (катионный липид), дипальмитоилфосфатидилхолин (DPPC; Avanti Polar Lipids), синтетический холестерин (Sigma) и ПЭГ-C-DMA в молярном отношении 57,1:7,1:34,3:1,4. После формирования нагруженных частиц SNALP были диализированы против PBS, фильтрат был простерилизован через 0,2 мкм-фильтр перед использованием. Средние размеры частиц составляли 75-85 нм, и 90-95% миРНК были инкапсулированы в липидных частицах. Окончательное отношение миРНК/липид в составах, использовавшихся для in vivo тестирования, составляло ~0,15 (вес/вес). Системы LNP-миРНК, содержавшие миРНК против фактора VII, были сразу растворены до соответствующих концентраций в стерильном PBS перед использованием, и составы были введены внутривенно через боковую хвостовую вену в суммарном объеме 10 мл/кг. Этот способ и эти системы доставки могут быть экстраполированы на нацеленную на нуклеиновую кислоту систему по настоящему изобретению.
[607] Другие липиды
[608] Другие катионные липиды, такие как аминолипид 2,2-дилинолеил-4-диметиламиноэтил-[1,3]-диоксолан (DLin-KC2-DMA) могут быть использованы, чтобы инкапсулировать нацеленную на нуклеиновую кислоту систему, компоненты этой системы или молекулу (молекулы) нуклеиновой кислоты, кодирующие компоненты этой системы, например, подобные миРНК (см., например, Jayaraman, Angew. Chem. Int. Ed. 2012, 51, 8529-8533) и, следовательно, могут использоваться в практике изобретения. Может быть рассмотрен предварительно сформированная везикула со следующим липидным составом: аминолипид, дистеароилфосфатидилхолин (DSPC), холестерин и (R)-2,3-бис(октадецилокси)пропил-1-(метоксиполи(этиленгликоль)2000)пропилкарбамат (ПЭГ-липид) в молярном отношении 40/10/40/10, соответственно, и отношении мРНК фактора свертывания крови VII (FVII)/общее количество липидов, приблизительно равном 0,05 (в весовом отношении). Чтобы гарантировать узкое распределение размеров частиц в диапазоне 70-90 нм и низкий индекс полидисперсности 0,11+0,04 (n=56), частицы могут быть экструдированы до трех раз через 80-нм мембраны до добавления направляющей РНК. Могут быть использованы частицы, содержащие сильнодействующий аминолипид 16, в котором молярное отношение четырех липидных компонентов равно 16, DSPC, холестерин и ПЭГ-липид (50/10/38,5/1,5), которое может быть далее оптимизировано, чтобы увеличить активность in vivo.
[609] Michael S D Kormann et al. ("Expression of therapeutic proteins after delivery of chemically modified mRNA in mice: Nature Biotechnology, Volume:29, Pages: 154-157 (2011)) описывают использование липидных оболочек для доставки РНК. Использование липидных оболочек также предпочтительно в данном изобретении.
[610] В другом варианте осуществления изобретения липиды могут быть смешаны с нацеленной на нуклеиновую кислоту системой по настоящему изобретению, ее компонентом(ами) или молекулой(ами) нуклеиновой(ых) кислот(ы), кодирующей таковой(ые), для образования липидных наночастиц (LNP). Липиды включают, без наложения ограничений, DLin-KC2- DMA4, C12-200 и колипиды дистероилфосфатидилхолина, холестерин и ПЭГ-DMG могут быть смешаны с нацеленной на РНК системой вместо миРНК (см., например, Novobrantseva, Molecular Therapy-Nucleic Acids (2012) I, e4; doi:10.1038/mtna.2011.3) с использованием процедуры спонтанного образования везикул. Молярное отношение компонентов может быть примерно 50/10/38,5/1,5 (DLin-KC2-DMA или C12-200/дистероилфосфатидилхолин/холестерин/ПЭГ-DMG). Конечное весовое соотношение липид:РНК может составлять 12:1 и 9:1 в случае липидных частиц (LNP) с DLin-KC2-DMA и C12-200, соответственно. Такие составы могут содержать частицы со средним диаметром ~80 нм при эффективности захвата >90%. Может предусматриваться доза 3 мг/кг.
[611] Tekmira располагает портфолио, содержащим примерно 95 семейств патентов в США и в других странах, относящихся к различным аспектам липидных наночастиц (LNP) и их смесей (см., например, патенты США №7982027; 7799565; 8058069; 8283333; 7901708; 7745651; 7803397; 8101741; 8188263; 7915399; 8236943 и 7838658 и европейские патенты №1766035; 1519714; 1781593 и 1664316), все могут быть использованы и/или адаптированы для использования в настоящем изобретении.
[612] Нацеленная на нуклеиновую кислоту система или ее компоненты или молекула(ы), кодирующая(ие) таковые, могут быть доставлены в упакованном в микросферы PLGA виде таким образом, как описано в опубликованных патентных публикациях США 20130252281, 20130245107 и 20130244279 (приписываются Modema Therapeutics), относящихся к аспектам составления композиций, включающих молекулы модифицированной нуклеиновой кислоты, которые могут кодировать белок, предшественник белка или частично или полностью процессированную форму белка или предшественника белка. Смесь может иметь молярное соотношение 50:10:38,5:1,5:3,0 (катионный липид: фузогенный липид:холестерин:ПЭГ-липид). ПЭГ-липид может быть выбран, но не ограничиваясь ими, из ПЭГ-c-DOMG, ПЭГ-DMG. Фузогенным липидом может являться DSPC. См. также Schrum et aJ., Delivery and Formulation of Engineered Nucleic Acids, опубликованное приложение US 20120251618.
[613] Технологии Nanomerics призваны преодолеть трудности, связанные с биодоступностью для широкого круга терапевтических применений, включая низкомолекулярные гидрофобные лекарства, пептиды и основанные на нуклеиновых кислотах препараты (плазмиды, миРНК, микроРНК). Конкретные пути введения, для которых показаны значительные преимущества данной технологии, включают пероральное введение, транспортировка через гематоэнцефалический барьер, доставка в солидные опухоли, а также глаз. См., например, Mazza et al., 2013, ACS Nano. 2013 Feb 26;7(2): 1016-26; Uchegbu and Slew, 2013, J Pharm Sci. 102(2):305-10 и Lalatsa et al., 2012, J Control Release, 2012 Jul 20; 161 (2):523-36.
[614] В публикации патентной заявки США №20050019923 описываны катионные дендримеры для доставки биоактивных молекул, таких как молекулы полинуклеотидов, пептидов или полипептидов, и/или фармакологических агентов, в тело млекопитающего. Такие дендримеры подходят для направления доставки биоактивных молекул, например, в печень, селезенку, легкие, почку или сердце (или даже в головной мозг). Дендримеры представляют собой трехмерные макромолекулы, полученные за несколько этапов из простых разветвленных единиц-мономеров, природа и функциональность которых может легко контролироваться и изменяться. Дендримеры синтезируют путем повторяющегося добавления структурных единиц к мультифункциональному ядру (дивергентный способ синтеза) или их наращивания в направлении мультифункционального ядра (конвергентный способ синтеза), и каждое добавление к трехмерной оболочке структурных единиц приводит к образованию следующего поколения дендримеров. Полипропилениминовые дендимеры образуются, начиная с диаминобутанового ядра, к которому присоединяются вдвое больше аминогруппп в реакции Михаэля с присоединением акрилонитрила к первичным аминам и последующей гидрогенизацией нитрилов. Это приводит к удвоению количества аминогрупп. Полипропилениминовые дендримеры содержат 100% протонируемых азотов и до 64 терминальных аминогрупп (поколение 5, DAB 64). Протонируемыми группами обычно являются аминогруппы, которые способны присоединять протоны при нейтральном pH. Использование дендримеров в качестве агентов доставки генов было в значительной степени сфокусировано на использовании полиамидоамина, и фосфорсодержащие соединения со смесью аминов/амидов или N-P(02)S как конъюгирующих единиц, соответственно, при этом не было сообщений об использовании предшествующих поколений полипропилениминовых дендримеров для доставки генов. Полипропилениминовые дендримеры также исследовались в качестве чувствительных к pH систем контролируемого высвобождения для доставки лекарств и для их упаковки в качестве гостевых молекул при их химической модификации периферическими аминокислотными группами. Цитотоксичность и взаимодействие полипропилениминовых дендримеров с ДНК, а также эффективность трансфекции DAB 64 также были изучены.
[615] Патентная публикация США №20050019923 основана на наблюдении о том, что, в противоречие с более ранними сообщениями, катионные дендримеры, такие как полипропилениминовые дендримеры, демонстрируют подходящие свойства, такие как специфическое нацеливание и низкая токсичность, для использования для направленной доставки биоактивных молекул, таких как молекулы генетического материала. В дополнение к этому, производные катионных дендримеров также демонстрируют подходящие свойства для направленной доставки биоактивных молекул. См. также Bioactive Polymers, опубликованная заявка США 20080267903, в которой описано: "Различные полимеры, включая катионные полиаминные полимеры и дендримерные полимеры, как было показано, обладают антипролиферативной активностью, и следовательно могут быть полезны для лечения заболеваний, характеризуемых нежелательной пролиферацией клеток, таких как новообразования и опухоли, воспалительные заболевания (включая аутоиммунные заболевания), псориаз и атеросклероз. Такие полимеры могут быть использованы как самостоятельно в качестве активных агентов или в качестве "контейнера" для доставки других терапевтических агентов, таких как молекулы лекарств или нуклеиновые кислоты для генной терапии. В таких случаях собственная присущая полимеру противоопухолевая активность может дополнять активность доставляемого агента. Утверждения из данной патентной публикации могут быть использованы в совокупности с приведенными в настоящем описании идеями для доставки нацеленной на нуклеиновую кислоту систем(ы) или ее компонента(ов), или кодирующую таковые молекулу(ы) нуклеиновой кислоты.
Сверхзаряженные белки
[616] Сверхзаряженные белки представляют собой класс полученных способами инженерии или природных белков с исключительно высоким положительным или отрицательным суммарным теоретическим зарядом, которые могут использоваться для доставки нацеленной на нуклеиновую кислоту системы(систем), или ее компонента(ов), или молекулы(молекул) нуклеиновой кислоты, кодирующей их. Несущие как сверхположительный, так и сверхотрицательный заряд белки демонстрируют выдающуюся устойчивость к вызванному термически или химически агрегированию. Сверхположительно заряженные белки также способны проникать в клетки млекопитающих. Связывание с этими белками груза, такого как ДНК плазмиды, РНК или другие белки, может сделать возможной функциональную доставку этих макромолекул в клетки млекопитающих in vitro и in vivo. Лаборатория под руководством David Liu сообщила о создании и описании сверхзаряженных белков в 2007 года (Lawrence et. al., 2007, Journal of the American Chemical Society 129, 10110-10112).
[617] Отличная от вирусной доставка РНК и ДНК плазмиды в клетки млекопитающих представляет ценность как для исследований, так и для терапевтических применений (Akinc et al., 2010, Nat. Biotech. 26, 561-569). Очищенный белок +36 GFP (или другой сверхположительно заряженный белок) смешивали с РНК на соответствующих питательных средах без сыворотки и оставляли для образования комплексов перед добавлением к клеткам. Добавление сыворотки на этой стадии ингибирует формирование сверхзаряженных комплексов белок-РНК и снижает эффективность лечения. Приведенный ниже протокол продемонстрировал эффективность для ряда клеточных линий (McNaughton et al., 2009, Proc. Natl. Acad. Sci, USA 106, 6111-6116). Однако экспериментальные эксперименты, изменяющие дозу белка и РНК, должны быть выполнены, чтобы оптимизировать процедуру специфичных клеточных линий. Однако следует провести пробные эксперименты с изменениями дозы белка и РНК для оптимизации использования процедуры применительно к конкретным клеточным линиям.
(1) За один день до обработки высевают 1×105 клеток на лунку в 48-луночный планшет.
(2) В день обработки растворяют очищенный белок +36 GFP в питательных средах без сыворотки до конечной концентрации 200 нМ. Добавляют РНК до конечной концентрации 50 нМ. Перемешивают при помощи устройства для встряхивания и инкубируют при комнатной температуре в течение 10 минут.
(3) Во время инкубации удаляют питательную среду клеток и промывают PBS один раз.
(4) После инкубации +36 GFP с РНК к клеткам добавляют комплексы белок-РНК.
(5) Инкубируют клетки с комплексами при 37°C в течение 4 часов.
(6) После инкубации отбирают питательные среды и промывают три раза раствором гепарина в PBS (20 Е/мл). Инкубируют клетки с содержащими сыворотку питательными средами на протяжении 48 ч или более в зависимости от анализа активности.
(7) Анализируют клетки при помощи иммуноблоттинга, к-ПЦР, анализа фенотипа или другого соответствующего способа.
[618] Лаборатория под руководством David Liu также установила, что белок +36 GFP является эффективным агентом доставки плазмид в различные клетки. Поскольку ДНК плазмиды является большим грузом, чем миРНК, для эффективного образования комплексов с плазмидами требуется пропорционально большее количество белков +36 GFP. Для эффективной доставки плазмиды заявители доставки разработали вариант +36 GFP, несущий C-концевую метку HA2, пептида, для которого известна способность разрушать эндосомы, полученного из белка гемагглютинина вируса гриппа. Следующий протокол продемонстрировал эффективность для ряда клеток, однако как и рекомендовано выше, необходимо, чтобы дозы ДНК плазмиды и сверхзаряженного белка были оптимизированы для конкретных клеточных линий и различных применений способа доставки.
(1) За день до обработки высевают по 1×105 клеток на лунку в 48-луночный планшет.
(2) В день обработки растворяют очищенный белок +36 GFP в питательных средах без сыворотки до конечной концентрации 2 нМ. Добавляют 1 мг ДНК плазмиды. Перемешивают с помощью устройства для встряхивания и инкубируют при комнатной температуре на протяжении 10 мин.
(3) Во время инкубации отбирают питательные среды клеток и промывают PBS один раз.
(4) После инкубации +36 GFP и ДНК плазмиды осторожно добавляют комплексы белок-ДНК к клеткам.
(5) Инкубируют клетки с комплексами при 37°C на протяжении 4 ч.
(6) После инкубации удаляют питательные среды, и промывают клетки PBS. Инкубируют клетки в содержащих сыворотку питательных средах и инкубируют следующие 24-48 часов.
(7) Анализируют доставку плазмиды (например, по управляемой плазмидой экспрессии генов) в зависимости от ситуации.
[619] См. также, например, McNaughton et al., Proc. Natl. Acad. Sci. USA 106, 6111-6116 (2009); Cronican et al., ACS Chemical Biology 5, 747-752 (2010); Cronican et al., Chemistry & Biology 18, 833-838 (2011); Thompson et al., Methods in Enzymology 503, 293-319 (2012); Thompson, D.B., et al., Chemistry & Biology 19 (7), 831-843 (2012). Способы с применением сверхзаряженных белков могут использоваться и/или адаптированы для доставки нацеленной на нуклеиновую кислоту системы по настоящему изобретению. Такие системы Dr. Lui и приведенные в настоящей заявке документы в совокупности с изложенными в настоящем описании идеями могут использоваться при доставке нацеленной на нуклеиновую кислоту систем(ы) или ее компонента(ов) или кодирующей молекул(ы) нуклеиновой кислот.
Пептиды, проникающие в клетки (CPP)
[620] В еще одном варианте осуществления изобретения рассмотрено использование пептидов, проникающих в клетки (CPP), для доставки системы CRISPR-Cas. CPP представляют собой короткие пептиды, которые облегчают захват клетками различных молекулярных грузов (от наноразмерных частиц до маленьких химических молекул и больших фрагментов ДНК). Термин "груз" как используют в настоящем описании, включает, но не ограничивается ими, состоящую из терапевтических агентов, диагностических зондов, пептидов, нуклеиновых кислот, антисмысловых олигонуклеотидов, плазмид, белков, частиц, включая наночастицы, липосом, хромофоров, низкомолекулярных соединений и радиоактивных материалов. Аспекты изобретения подразумевают также, что груз может также включать любой компонент системы CRISPR-Cas или целую функциональную систему CRISPR-Cas. В одном аспекте изобретение относится к следующим способам доставки желаемого груза индивиду, включающим: (a) получение комплекса, включающего пептид, который способен проникать в клетки, и желаемый груз, и (b) пероральное, внутрисуставное, внутрибрюшинное, интратекальное, внутриартериальное, интраназальное, внутрипаренхимное, подкожное, внутримышечное, внутривенное, дермальное, ректальное или местное введение комплекса индивиду. Груз связывают с пептидами посредством химической связи (ковалентной) или посредством нековалентных взаимодействий.
[621] Функцией проникающих в клетки пептидов (CPP) является доставка груза в клетки - процесс, который обычно осуществляется посредством эндоцитоза груза, доставленного к эндосомам живых клеток млекопитающих. Проникающие в клетку пептиды (CPP) имеют различные размеры, последовательность аминокислот и заряд, но все CPP объединяет одна отличительная особенность, которая состоит в способности перемещаться через плазменную мембрану и облегчать доставку различных молекулярных грузов в цитоплазму или органеллы. Такое перемещение CPP может быть разделено на три главные группы в соответствии с механизмами входа: прямое проникновение через мембрану, опосредованный эндоцитозом вход и перемещение с формированием переходной структуры. CPP нашли многочисленные способы применения в медицине в качестве агентов доставки лекарственных средств при лечении различных заболеваний, включая противораковые средства и средства для лечения вирусных заболеваний, а также контрастирующих агентов для маркировки клетки. Примеры последних включают использование в качестве переносчика GFP, контрастирующих агентов для МРТ или квантовых точек. CPP имеют большой потенциал в качестве векторов как in vitro, так и in vivo доставки для исследований и использования в медицине. CPP, как правило, либо имеют аминокислотный состав с высоким относительным содержанием положительно заряженных аминокислот, таких как лизин или аргинин, либо содержат последовательности, имеющие чередующийся паттерн полярных/заряженных аминокислот и неполярных, гидрофобных аминокислот. Эти два типа структур обозначают как поликатионные или амфифильные соответственно. Третий класс CPP представляет собой гидрофобные пептиды, содержащие только неполярные остатки с низким суммарным зарядом или имеющие гидрофобные аминокислотные группы, которые крайне важны для клеточного захвата. Один из первых обнаруженных CPP - трансактивирующий активатор транскрипции (Tat) вируса иммунодефицита человека 1 (ВИЧ-1), который эффективно захватывается из питательных сред многочисленными типами культивируемых клеток. Позднее количество известных CPP значительно увеличилось, и были получены небольшие молекулярные синтетические аналоги, более эффективные при трансдукции белка. CPP включают, но не ограничиваются ими: пенетратин, Tat (48-60), транспортан и (R-AhX-R4) (Ahx=аминогексаноил).
[622] В патенте США 8372951 описан CPP, полученный из эозинофильного катионного белка (ECP), который демонстрирует высокую эффективность проникания в клетку и низкую токсичность. Также описаны особенности доставки CPP с грузом в представителях позвоночных. Будущие аспекты CPP и их доставки описаны в патентах США 8575305; 8614194 и 8044019. CPP может использоваться для доставки системы CRISPR-Cas или ее компонентов. Использование CPP для доставки системы CRISPR-Cas или ее компонентов также описано в рукописи "Gene disruption by cell-penetrating peptide-mediated delivery of Cas9 protein and guide RNA", by Suresh Ramakrishna, Abu-Bonsrah Kwaku Dad, Jagadish Beloor, et al. Genome Res. 2014 Apr 2, [Epub в печати], включенной в настоящее описание в качестве ссылки в полном оъеме, где продемонстрировано, что лечение рекомбинантным белком Cas9, конъюгированным с CPP, и направляющими РНК в комплексе с CPP приводит к генным нарушениям эндогенного характера в клеточных линиях человека. В статье белок Cas9 был связан с CPP через тиоэфирную связь, тогда как направляющая РНК была в комплексе с CPP, формируя уплотненные, положительно заряженные частицы. Было показано, что одновременная и последовательная обработка клеток человека, включая эмбриональные стволовые клетки, фибробласты кожи, клетки HEK293T, клетки HeLa и эмбриональные клетки карциномы, модифицированным Cas9 и направляющей РНК, ведет к эффективным генным изменениям с уменьшением количества нецелевых мутаций по сравнению с трансфекцией плазмидами.
Имплантируемые устройства
[623] В другом варианте осуществления изобретения имплантируемые устройства также предусматриваются для доставки нацеленной на нуклеиновую кислоту системы, ее компонента (компонентов) или молекулы (молекул) нуклеиновых кислот, кодирующих их. Например, в публикации патентной заявки США 20110195123 описано имплантируемое медицинское устройство, которое местно элюирует лекарственное средство и находится в человеке длительное время, включая несколько типов такого устройства, способы лечения и способы имплантации. Устройство, включающее полимерную подложку, такую как матрица, например, которая используется в качестве корпуса устройства, и лекарственное средство, и в некоторых случаях дополнительные каркасные материалы, такие как металлы или дополнительные полимеры и материалы, чтобы увеличить видимость и визуализацию. Имплантируемое устройство может быть предпочтительно для местной доставки лекарственного средства в течение длительного периода, когда препарат доставляется непосредственно во внеклеточный матрикс (ECM) пораженной заболеванием области, такой как опухоль, область воспаления, дегенерации, или для симптоматического лечения, или к травмированным клеткам гладкой мускулатуры, или для профилактики. Одним типом лекарственного средства является РНК, как описано выше, и эта система может быть использована/и или адаптирована для нацеленной на нуклеиновую кислоту системы по настоящему изобретению. Способы имплантации в некоторых вариантах осуществления изобретения представляют собой существующие способы имплантации, которые разработаны и используются в настоящее время для других способов лечения, включая брахитерапию и биопсию при помощи иглы. В таких случаях размеры нового импланта, описанного в этом изобретении, подобны оригинальному импланту. Как правило, несколько устройств могут быть имплантированы во время одной лечебной процедуры.
[624] В публикации патента США 20110195123 описано лекарственное средство, которое обеспечивает имплантируемую или вставную систему, включая системы, применимые для полостей, таких как брюшная полость, и/или любого другого типа введения, в которых система доставки лекарственного средства не закреплена или прикреплена, содержащую биостабильную и/или разлагаемую и/или биоабсорбируемую полимерную подложку, которая в некоторых случаях может представлять собой, например, матрицу. Следует отметить, что термин "установка" также включает имплантацию. Система доставки лекарственного средства предпочтительно реализуется как "Loder", как описано в публикации патента США 20110195123.
[625] Полимер или множество полимеров являются биосовместимыми, включающими агент и/или множество агентов, что позволяет высвобождать агент с контролируемой скоростью, причем общий объем полимерного субстрата, такого как матрица, например, в некоторых вариантах осуществления необязательно и предпочтительно не превышают максимального объема, который позволяет достичь терапевтического уровня агента. В качестве неограничивающего примера такой объем предпочтительно находится в диапазоне от 0,1 м3 до 1000 мм3, как того требует объем для загрузки агента. "Loder" в некоторых случаях может быть более крупным, например, когда он включен в устройство, размер которого определяется функциональностью, например, но без ограничения, коленный сустав, кольцо шейки матки и тому подобное.
[626] Система доставки лекарственного средства (для доставки состава) сконструирована в некоторых вариантах осуществления, чтобы предпочтительно использовать разлагаемые полимеры, где основным механизмом высвобождения является объемная эрозия; или в некоторых вариантах осуществления используются нерастворимые или медленно деградирующие полимеры, в которых основным механизмом высвобождения является диффузия, а не объемная эрозия, так что наружная часть функционирует как мембрана, а ее внутренняя часть функционирует как резервуар для лекарственных средств, который практически не испытывает влияния окружения в течение длительного периода времени (например, от недели до нескольких месяцев). Могут также использоваться комбинации различных полимеров с различными механизмами высвобождения. Градиент концентрации на поверхности предпочтительно эффективно поддерживается постоянным в течение значительной части полного периода высвобождения лекарственного средства, и поэтому скорость диффузии является фактически постоянной (называемой диффузией "нулевого режима"). Под термином "постоянная" подразумевается скорость диффузии, которая предпочтительно поддерживается выше нижнего порога терапевтической эффективности, но которая может по-прежнему в некоторых случаях иметь начальный всплеск и/или может колебаться, например увеличиваясь и уменьшаясь в определенной степени. Скорость диффузии предпочтительно поддерживают в течение длительного периода времени, и ее можно считать постоянной до определенного уровня для оптимизации терапевтически эффективного периода, например эффективного периода подавления экспрессии генов.
[627] Система доставки лекарственного средства необязательно и предпочтительно предназначена для защиты терапевтического агента на основе нуклеотидов от деградации, будь то химическая по своей природе или из-за действия ферментов и других факторов в организме индивида.
[628] Система доставки лекарственного средства, описанная в публикации патента США 20110195123, в некоторых случаях связана с чувствительными и/или активирующими устройствами, которые работают во время и/или после имплантации устройства, посредством неинвазивных и/или минимально инвазивных способов активации и/или ускорения/замедления, например, необязательно включая, но не ограничиваясь ими, нагревание и охлаждение, лазерные лучи и ультразвук, включая фокусированные ультразвуковые и/или радиочастотные (RF) способы или устройства.
[629] Согласно некоторым вариантам публикации патента США 010195123, область для местной доставки может необязательно включать целевые области, характеризующиеся высокой аномальной пролиферацией клеток и подавленным апоптозом, включая опухоли, активные и хронические воспаления и инфекции, включая аутоиммунные заболевания, дегенерацию тканей, включая мышцы и нервную ткань, хронические боли, участки дегенерации и места переломов костей и других ран для улучшения регенерации ткани и поврежденной сердечной, гладкой и поперечно-полосатой мускулатуры.
[630] Область для имплантации композиции или целевая область, предпочтительно представляет собой радиус, область и/или объем, который достаточно мал, для целенаправленной локальной доставки. Например, целевой участок произвольно имеет диаметр в диапазоне приблизительно от 0,1 мм до приблизительно 5 см.
[631] Местоположение целевой области должно быть выбрано предпочтительно для максимальной терапевтической эффективности. Например, композицию системы доставки лекарственных средств (необязательно с устройством для имплантации, как описано выше) необязательно и предпочтительно имплантируют непосредственно в саму опухоль или ее ближайшее окружение или связанные с ней кровеносные сосуды.
[632] Например, композицию (необязательно вместе с устройством) необязательно имплантируют непосредственно в или вблизи поджелудочной железы, предстательной железы, молочных желез, печени, через сосок, в сосудистую систему и т.д.
[633] Положение целевой области может быть выбрано из группы, включающей, по существу состоящей или состоящей из следующих примеров (не налагающих ограничений, поскольку любой участок тела может подходить для имплантации Loder): 1. дегенеративные участки мозга в базальных ганглиях, белом и сером веществе при заболеваниях Паркинсона или Альцгеймера, 2. позвоночник, например, в случае бокового амиотрофического склероза (ALS); 3. шейка матки для предотвращения инфекции вирусом папилломы человека (HPV); 4. суставы с острым и хроническим воспалением; 5. кожа, например, в случае псориаза; 6. симпатические и сенсорные нервы для получения обезболивающего эффекта; 7. внутрикостная имплантация; 8. области острой и хронической инфекции; 9. внутривлагалищная имплантация; 10. внутреннее ухо - слуховая система, лабиринт внутреннего уха, вестибулярная система; 11. внутритрахеальная имплантация; 12. внутрисердечная, коронарная, эпикардиальная имплантация; 13. имплантация в мочевой пузырь; 14. имплантация в желчную систему; 15. паренхимная ткань, включающая, не ограничиваясь, почкой, печенью, селезенкой; 16. лимфатические узлы; 17. слюнные железы; 18. десны; 19. внутрисуставная имплантация (в суставы); 20. внутриглазная имплантация; 21. ткани головного мозга; 22. желудочки мозга; 23. полости, включая брюшную полость (включая, не ограничиваясь, рак яичника); 24. внутрь пищевода и 25. ректально.
[634] В некоторых случаях введение системы (например, устройства, содержащего состав), связано с инъекцией материала во внеклеточный матрикс в целевой области и вблизи этой области для изменения локального значения pH, и/или температуры, и/или других биологических факторов, влияющих на диффузию препарата и/или фармакокинетику во внеклеточном матриксе целевой области и вблизи нее.
[635] В некоторых случаях, согласно некоторым вариантам осуществления изобретения, высвобождение указанного агента может быть связано с сенсорами и/или активирующими устройствами, управление которыми производится до и/или во время и/или после имплантации, неинвазивными и/или минимально инвазивными и/или другими способами активации и/или способами ускорения/замедления, включая использование лазерных лучей, радиации, нагревания и охлаждения, и ультразвуковое воздействие, включая фокусированный ультразвук и/или радиочастотные способы или устройства, а также химические активаторы.
[636] Согласно другим вариантам осуществления изобретения, описанным в публикации патента США 20110195123, используемый препарат предпочтительно включает РНК, например для локализованных случаев рака молочной железы, поджелудочной железы, мозга, почки, мочевого пузыря, легкого и простаты, как описано ниже. Несмотря на то, что в качестве примера приводится РНК-интерференция, многие лекарственные препараты применимы при инкапсулирования в "Loder", и могут использоваться совместно с данным изобретением при условии, что такие лекарственные препараты сами по себе могут быть инкапсулированы в субстрат "Loder", например, такой как матрица, и эта система может быть использована и/или адаптирована для доставки нацеленной на нуклеиновую кислоту системы по настоящему изобретению.
[637] В качестве другого примера конкретного применения можно привести нервные и мышечные дегенеративные заболевания, развивающиеся из-за аномальной экспрессии генов. Локальная доставка РНК может проявлять терапевтическое действие свойства для изменения аномальной экспрессии генов. Локальная доставка антиапоптотических, противовоспалительных и антидегенеративных лекарственных препаратов, включая небольшие по размеру молекулы препаратов и макромолекулы может также быть терапевтической. В таких случаях "Loder "применяется для длительного высвобождения с постоянной скоростью и/или через специализированное устройство, которое имплантируется отдельно. Все это может быть использовано и/или адаптировано для нацеленной на нуклеиновую кислоту системы по настоящему изобретению.
[638] В качестве еще одного примера конкретного применения могут быть приведены психиатрические и когнитивные заболевания, которые можно лечить с помощью модификаторов активности генов. Генный нокдаун является вариантом лечения этих заболеваний. "Loder", местно доставляющие терапевтические агенты к участкам центральной нервной системы, являются терапевтическими вариантами лечения психиатрических и когнитивных заболеваний включая, но не ограничиваясь ими, психоз, биполярные расстройства, невротические заболевания и поведенческие расстройства. "Loder" также могут местно доставлять лекарственные препараты, включая небольшие молекулы лекарственных препаратов и макромолекулы после имплантации в конкретные области головного мозга. Все это может быть использовано и/или адаптировано для нацеленной на нуклеиновую кислоту системы по настоящему изобретению.
[639] Другим примером конкретного применения является подавление экспрессии врожденных и/или адаптивных медиаторов иммунитета в локальных участках, что позволяет предотвратить отторжение трансплантатов. Локальная доставка РНК и иммуномодулирующих реактивов с использованием Loder, имплантированного в пересаженный орган и/или область имплантации, способствует локальному иммунному подавлению, отражая иммуноциты, такие как CD, активированные в отношении пересаженного органа. Все это может быть использовано/и или адаптировано для нацеленной на нуклеиновую кислоту системы по настоящему изобретению.
[640] В качестве другого примера конкретного применения можно привести факторы роста сосудов, включая VEGF, ангиогенин и другие, необходимые для неоваскуляризации. Локальная доставка факторов, пептидов, пептидомиметиков или подавление их репрессоров является важным терапевтическим способом; подавление репрессоров и локальная доставка факторов, пептидов, макромолекул и небольших молекул лекарств, стимулирующих ангиогенез с помощью "Loder", являются терапевтическими для периферических, системных и сердечно-сосудистых заболеваний.
[641] Способ введения, такой как имплантация, может уже использоваться для других типов имплантации тканей и/или для введения и/или для отбора проб тканей, в некоторых случаях без модификаций, или, альтернативно, в некоторых случаях только с незначительными модификациями в таких способах. Такие способы включают, но не ограничиваются ими, способы брахитерапии, биопсии, эндоскопии вместе с/или без применения ультразвука, такие как ретроградная холангиопанкреатография (ERCP,) стереотаксические способы для тканей головного мозга, лапароскопию, включая имплантацию лапароскопом в суставы, органы брюшной полости, стенку мочевого пузыря и полости тела.
[642] Обсуждаемая в настоящем описании технология имплантируемых устройств может быть использована в рамках настоящего изобретения и, следовательно, благодаря этой информации и знаниям в данной области, система CRISPR-Cas, ее компоненты, их молекулы нуклеиновых кислот, или кодирующие или обеспечивающие их компоненты могут быть доставлены через имплантируемое устройство.
Пациент-специфические способы скрининга
[643] Нацеленная на нуклеиновую кислоту система, которая нацелена на РНК, например, тринуклеотидные повторы, может быть использована для скрининга пациентов или образцов клеток/тканей пациентов на предмет наличия таких повторов. Повторы могут быть мишенью РНК системы нацеливания на нуклеиновую кислоту, и при наличии связывания с ней с помощью системы нацеливания на нуклеиновую кислоту, это связывание может быть обнаружено, тем самым указывая на то, что такой повтор присутствует. Таким образом, систему нацеливания на нуклеиновую кислоту можно использовать для скрининга пациентов или образцов клеток/тканей пациентов на наличие повторов. Затем пациенту можно назначить подходящее соединение (соединения) для лечения этого состояния; или может быть введена система нацеливания на нуклеиновую кислоту для связывания, которое приводит к инсерции, делеции или мутации и облегчению состояния.
[644] Изобретение относится к нуклеиновым кислотам для связывания последовательностей РНК-мишеней.
мРНК эффекторного белка CRISPR и направляющая РНК
[645] мРНК эффекторного белка CRISPR и направляющая РНК могут также доставляться отдельно. мРНК эффекторного белка системы CRISPR можно доставить до направляющей РНК, чтобы дать время для экспрессии белка CRISPR. мРНК эффекторного белка системы CRISPR можно вводить за 1-12 часов (предпочтительно приблизительно 2-6 часов) до введения направляющей РНК.
[646] Альтернативно, мРНК эффекторного белка CRISPR и направляющую РНК можно вводить вместе. Предпочтительно вторую усиливающую дозу направляющей РНК можно вводить через 1-12 часов (предпочтительно приблизительно 2-6 часов) после первоначального введения мРНК эффекторного белка CRISPR+направляющей РНК.
[647] Эффекторный белок CRISPR согласно настоящему изобретению, а именно эффекторный белок C2c2, иногда упоминается в настоящем описании как фермент CRISPR. Понятно, что эффекторный белок основан или получен из фермента, поэтому термин "эффекторный белок", безусловно, включает "фермент" в некоторых вариантах осуществления изобретения. Однако также следует понимать, что эффекторный белок может, как требуется в некоторых вариантах осуществления, связывать ДНК или РНК, но не обязательно разрезать или вносить одноцепочечный разрыв, что является функцией "мертвого" эффекторного белка Cas.
[648] Дополнительное введение мРНК эффекторного белка CRISPR и/или направляющей РНК может быть полезным для достижения наиболее эффективных уровней модификации генома. В некоторых вариантах осуществления фенотипическое изменение предпочтительно является результатом модификации генома, когда мишенью является генетическое заболевание, особенно в способах терапии, и предпочтительно, когда обеспечивается матрица репарации для коррекции или изменения фенотипа.
[649] В некоторых вариантах осуществления заболевания, которые могут быть мишенью, включают заболевания, которые связаны с дефектами сплайсинга.
[650] В некоторых вариантах осуществления клеточные мишени включают гемопоэтические стволовые клетки/клетки-предшественники (CD34+); Т-клетки человека; и клетки сетчатки - например, клетки-предшественники фоторецепторов.
[651] В некоторых вариантах осуществления гены-мишени включают: ген бета-глобина человека - HBB (для лечения серповидноклеточной анемии, включая стимуляцию генной конверсии (с использованием близкородственного гена HBD в качестве эндогенной матрицы)); CD3 (Т-клетки) и CEP920 - сетчатка (глаза).
[652] В некоторых вариантах осуществления болезни-мишени также включают: злокачественную опхуоль; серповидноклеточную анемию (основанную на точечной мутации); ВИЧ; бета-талассемию; и офтальмологические или глазные заболевания - например, амавроз Лебера (LCA), вызываемый дефектом сплайсинга.
[653] В некоторых вариантах осуществления способы доставки включают: опосредованную катионными липидами "прямую" доставку комплекса фермент-гид (рибонуклеопротеин) и электропорацию плазмидной ДНК.
[654] Способы по изобретению могут дополнительно включать доставку матриц, таких как матрицы репарации, которые могут представлять собой дцОДН или оцОДН, см. ниже. Доставка матриц может быть произведена одновременно или отдельно от доставки любого или всех эффекторных белков CRISPR или направляющих молекул, и через один и тот же механизм доставки или любой другой. В некоторых вариантах осуществления предпочтительно, чтобы матрица доставлялась вместе с направляющей молекулой и также предпочтительно эффекторным белком CRISPR. Примером может служить вектор AAV.
[655] Способы по изобретению могут дополнительно включать: (а) доставку в клетку двухцепочечного олигодезоксинуклеотида (дцОДН), включающего выступающие концы, комплементарные выступающим концам, созданным упомянутым разрывом двойной цепи, причем указанный дцОДН встраивается в локус-мишень, или - (б) доставку в клетку одноцепочечного олигодезоксинуклеотида (оцОДН), где указанный оцОДН действует как матрица для гомологичной направленной репарации разрыва двойной цепи. Способы по изобретению могут быть предназначены для профилактики или лечения заболевания у индивида, когда указанное заболевание вызвано дефектом в указанном локусе-мишени. Способы по изобретению могут осуществляться in vivo в индивидууме или ex vivo на клетке, взятой у индивида, где в некоторых случаях указанную клетку возвращают индивиду.
[656] Для минимизации токсичности и неспецифических эффектов важно контролировать концентрацию доставляемых мРНК эффекторного белка CRISPR и направляющей РНК. Оптимальные концентрации мРНК эффекторного белка CRISPR и направляющей РНК могут быть определены путем тестирования различных концентраций на клеточной или животной модели и с использованием глубокого секвенирования для анализа степени модификации в потенциально нецелевых геномных локусах. Например, для направляющей последовательности, нацеленной на 5'-GAGTCCGAGCAGAAGAAGAA-3' в гене EMX1 генома человека, глубокое секвенирование может быть использовано для оценки уровня модификации в следующих двух нецелевых локусах: 1: 5'-GAGTCCTAGC AGGAGAAG AA-3' и 2: 5'-GAGTCTAAGCAGAAGAAGAA-3'. Концентрация, которая дает наивысший уровень модификации мишени, минимизируя уровень внецелевой модификации, должна быть выбрана для доставки in vivo.
Индуцируемые системы
[657] В некоторых вариантах осуществления изобретения эффекторный белок CRISPR может составлять компонент индуцируемой системы. Индуцируемый характер системы позволяет пространственно-временный контроль редактирования генов или экспрессии генов с использованием энергии. Форма энергии может включать, но не ограничивается ими, электромагнитное излучение, звуковую энергию, химическую энергию и тепловую энергию. Примеры индуцируемой системы включают стимулирующие тетрациклин промоторы (Tet-On или Tet-Off), системы двугибридной активации транскрипции с малой молекулой (FKBP, ABA и т.д.) или светоиндуцируемые системы (фитохром, LOV-домены или криптохром). В одном варианте осуществления эффекторный белок CRISPR может быть частью светоиндуцируемого эффектора транскрипции (LITE) для прямого изменения транскрипционной активности последовательность-специфическим образом. Светоиндуцируемые компоненты могут включать эффекторный белок CRISPR, светочувствительный гетеродимер цитохрома (например, Arabidopsis thaliana) и домен активации/репрессии транскрипции. Другие примеры индуцибельных связывающих ДНК белков и способы их применения приведены в US 61/736465 и US 61/721283 и WO 2014018423 A2, которые включены в настоящее описание в качестве ссылки в полном объеме.
Иллюстративные способы применения системы CRISPR Cas
[658] Изобретение относится к не встречающейся в природе или сконструированной способами инженерии композиции, или одному или более полинуклеотидам, кодирующим компоненты указанной композиции, или векторам или системам доставки, содержащим один или более полинуклеотидов, кодирующих компоненты указанной композиции, для применения для модификации клетки-мишени in vivo, ex vivo или in vitro, и это может быть осуществлено способом, который изменяет клетку таким образом, что после модификации потомки или клеточная линия модифицированной клетки CRISPR сохраняет измененный фенотип. Модифицированные клетки и их потомки могут быть частью многоклеточного организма, такого как растение или животное, с применением системы CRISPR ex vivo к желаемым типам клеток. Изобретение CRISPR может представлять собой терапевтический способ лечения. Терапевтический способ лечения может включать редактирование гена или генома, или генную терапию.
[659] В одном аспекте изобретение относится к способам модификации полинуклеотида-мишени в эукариотической клетке, которая может быть in vivo, ex vivo или in vitro. В некоторых вариантах осуществления способ включает отбор образца клетки или популяции клеток у человека или животного, не являющегося человеком, и модификацию клетки или клеток. Культивирование может происходить на любом этапе ex vivo. Клетка или клетки могут быть повторно введены в животное или растение, отличное от человека. Для вновь введенных клеток особенно предпочтительно, чтобы клетки были стволовыми клетками.
[660] В некоторых вариантах осуществления способ включает предоставление возможности CRISPR-комплексу связываться с полинуклеотидом-мишенью для осуществления расщепления указанного полинуклеотида-мишени, в результате чего происходит модификация полинуклеотида-мишени, где комплекс CRISPR содержит эффекторный белок CRISPR и направляющую последовательность, гибридизованную или способную к гибридизации с последовательностью-мишенью в указанном полинуклеотиде-мишени.
[661] В одном из вариантов изобретение относится к способу модификации экспрессии полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления способ включает предоставление комплексу CRISPR возможности связываться с полинуклеотидом, так что указанное связывание приводит к увеличению или уменьшению экспрессии указанного полинуклеотида; где комплекс CRISPR содержит эффекторный белок CRISPR, объединенный с направляющей последовательностью, гибридизованной или способной к гибридизации с последовательностью-мишенью в указанном полинуклеотиде. Аналогичные соображения и условия применяются, как указано выше, для способов модификации полинуклеотида-мишени. Фактически, эти варианты отбора проб, культивирования и повторного введения применяются во всех аспектах настоящего изобретения.
[662] Действительно, в любом аспекте изобретения комплекс CRISPR может содержать эффекторный белок CRISPR в комплексе с направляющей последовательностью, гибридизованной или способной к гибридизации с последовательностью-мишенью. Аналогичные соображения и условия применяются, как указано выше, для способов модификации полинуклеотида-мишени.
[663] Таким образом, любой из не встречающихся в природе эффекторных белков CRISPR, описанных в настоящем описании, имеет по меньшей мере одну модификацию, и, тем самым, эффекторный белок обладает определенными улучшенными возможностями. В частности, любой из эффекторных белков способен образовывать комплекс CRISPR с направляющей РНК. Когда такой комплекс образуется, направляющая РНК способна связываться с полинуклеотидной последовательностью-мишенью, и эффекторный белок способен модифицировать локус-мишень. Кроме того, эффекторный белок в комплексе CRISPR уменьшает способность модифицировать один или более локусов-мишеней по сравнению с немодифицированным ферментом/эффекторным белком.
[664] Кроме того, модифицированные ферменты CRISPR, описанные в настоящем описании, охватывают ферменты которые обеспечивают повышение способности эффекторного белка комплекса CRISPR модифицировать один или более локусов-мишеней по сравнению с немодифицированным ферментом/эффекторным белком. Такая функция может быть предоставлена отдельно или предоставлена в сочетании с вышеописанной уменьшенной способностью модифицировать один или более локусов, не являющихся мишенями. Любые такие эффекторные белки могут быть снабжены любой из дополнительных модификаций в эффекторном белке CRISPR, как описано в настоящем описании, например, в сочетании с любой активностью, обеспечиваемой одним или более связанными гетерологичными функциональными доменами, любыми другими мутациями для снижения активности нуклеазы и т.п.
[665] В предпочтительных вариантах осуществления изобретения модифицированный эффекторный белок CRISPR обладает сниженной способностью модифицировать один или более локусов, не являющихся мишенями, по сравнению с немодифицированным ферментом/эффекторным белком и повышает способность модифицировать один или более локусов-мишеней по сравнению с немодифицированным ферментом/эффекторным белком. В сочетании с дополнительными модификациями эффекторного белка может быть достигнута значительно увеличенная специфичность. Например, комбинация таких предпочтительных вариантов осуществления с одной или более дополнительными мутациями предусматривается в том случае, если одна или более дополнительных мутаций находятся в одном или более активных каталитически активных доменах. В таких эффекторных белках повышенная специфичность может быть достигнута благодаря улучшенной специфичности в отношении активности эффекторного белка.
[666] Модификации для уменьшения нецелевых эффектов и/или усиления целевых эффектов, как описано выше, могут быть сделаны для аминокислотных остатков, расположенных в положительно заряженной области/желобке, расположенных между доменами RuvC-III и HNH. Понятно, что любой из описанных выше функциональных эффектов может быть достигнут путем модификации аминокислот в вышеупомянутом желобке, а также путем модификации аминокислот, смежных с этим желобком или вне него.
[667] Дополнительные функциональные возможности, которые могут быть внесены способами инженерии в модифицированные эффекторные белки CRISPR, как описано в настоящем описании, включают следующие. 1. модифицированные эффекторные белки CRISPR, которые нарушают взаимодействия ДНК:белок, не влияя на третичную или вторичную структуру белка. Это включает остатки, которые контактируют с любой частью дуплекса РНК:ДНК. 2. модифицированные эффекторные белки CRISPR, которые ослабляют внутрибелковые взаимодействия путем поддержания C2c2 в конформации, необходимой для нуклеазного разрезания в ответ на связывание ДНК (целевое или нецелевое). Например, модификация, которая мягко ингибирует, но все равно допускает конформацию нуклеазы домена HNH (расположена на расщепляющемся фосфате), 3. модифицированные эффекторные белки CRISPR, которые усиливают внутрибелковые взаимодействия путем поддержания C2c2 в конформации, ингибирующей активность нуклеазы в реакции связывания ДНК (целевой или нецелевой). Например: модификация, которая стабилизирует HNH-домен в конформации расщепляющегося фосфата. Любое такое дополнительное функциональное усиление может быть обеспечено в сочетании с любой другой модификацией эффекторного белка CRISPR, как подробно описано в данной заявке в другом пункте.
[668] Любая из описанных в настоящем описании улучшенных функциональных возможностей может быть внесена способами инженерии в любой эффекторный белок CRISPR, такой как эффекторный белок C2c2. Однако будет понятно, что любая из описанных в настоящем описании функциональных возможностей может быть внесена способами инженерии в эффекторные белки C2c2 из других ортологов, включая химерные эффекторные белки, содержащие фрагменты из множества ортологов.
[669] В рамках изобретения используются нуклеиновые кислоты для связывания последовательностей ДНК-мишеней. Это перспективно, так как нуклеиновые кислоты намного легче и дешевле производить, чем белки, а также специфичность может варьироваться в зависимости от длины участка, гомологии которого требуется. Например, не требуется сложное трехмерное позиционирование нескольких пальцев. Термины "полинуклеотид", "нуклеотид", "нуклеотидная последовательность", "нуклеиновая кислота" и "олигонуклеотид" используются взаимозаменяемо. Они относятся к полимерной форме нуклеотидов любой длины, либо дезоксирибонуклеотидов, либо рибонуклеотидов, или их аналогов. Полинуклеотиды могут иметь любую трехмерную структуру и могут выполнять любую из возможных известных или неизвестных функций. Ниже приведены неограничивающие примеры полинуклеотидов: кодирующие или некодирующие области гена или фрагмента гена, локусы (локус), определенные из анализа сцепления, экзоны, интроны, информационные РНК (мРНК), транспортные РНК (тРНК), рибосомные РНК (рРНК), малые интерферирующие РНК (миРНК), короткие шпилечные РНК (кшРНК), микроРНК, рибозимы, кДНК, рекомбинантные полинуклеотиды, разветвленные полинуклеотиды, плазмиды, векторы, выделенная ДНК любой последовательности, выделенная РНК любой последовательности, зонды нуклеиновых кислот и праймеры. Этот термин также охватывает структуры, подобные нуклеиновой кислоте, с синтетическими скелетами, см., например, Eckstein, 1991; Baserga et al., 1992; Milligan, 1993; WO 97/03211; WO 96/39154; Mata, 1997; Strauss-Soukup, 1997; and Samstag, 1996. Полинуклеотид может содержать один или более модифицированных нуклеотидов, таких как метилированные нуклеотиды и аналоги нуклеотидов. При их наличии, модификации нуклеотидной структуры могут происходить до или после сборки полимера. Последовательность нуклеотидов может быть прервана ненуклеотидными компонентами. Полинуклеотид может быть дополнительно модифицирован после полимеризации, например, путем конъюгации с меченым компонентом. Используемый в настоящем описании термин "дикий тип" является термином, понятным специалистам, и означает типичную форму организма, штамма, гена или характеристик, которая существует в природе, в отличие от мутантных или вариантных форм. "Дикий тип" может быть базовой линией. Как используют в настоящем описании, термин "вариант" следует понимать как проявление качеств, отличающихся от существующих в природе. Термины "не встречающиеся в природе" или "сконструированные способами инженерии" используются взаимозаменяемо и указывают на участие руки человека. Термины, относящиеся к молекулам нуклеиновой кислоты или полипептидам, означают, что молекула нуклеиновой кислоты или полипептид по меньшей мере по существу свободна от по меньшей мере одного другого компонента, с которым они естественно связаны в природе и существуют в природе. "Комплементарность" относится к способности нуклеиновой кислоты образовывать водородную связь (связи) с другой последовательностью нуклеиновой кислоты либо традиционным спариванием оснований Уотсона-Крика, либо другими нетрадиционными типами. Процентная комплементарность обозначает процентное содержание остатков в молекуле нуклеиновой кислоты, которые могут образовывать водородные связи (например, уотсон-криковское спаривание оснований) со второй последовательностью нуклеиновой кислоты (например, 5, 6, 7, 8, 9, 10 из 10 50%, 60%, 70%, 80%, 90% и 100% комплементарные). "Полностью комплементарные" означает, что все смежные остатки последовательности нуклеиновой кислоты связаны водородной связью с таким же количеством смежных остатков во второй последовательности нуклеиновой кислоты. "По существу комплементарные", как используется в настоящем описании, относится к степени комплементарности, которая составляет по меньшей мере 60%, 65%, 70%, 75%, 80%, 85%, 90%, 95%, 97%, 98%, 99% или 100% в области 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 30, 35, 40, 45, 50 или более нуклеотидов или относится к двум нуклеиновым кислотам, которые гибридизуются в жестких условиях. Как используют в настоящем описании, термин "жесткий условия" для гибридизации относится к условиям, при которых нуклеиновая кислота, имеющая комплементарность с последовательностью-мишенью, преимущественно гибридизуется с последовательностью-мишенью и по существу не гибридизуется с последовательностями, не являющимися мишенями. Жесткие условия, как правило, зависят от последовательности и меняются в зависимости от ряда факторов. В общем, чем длиннее последовательность, тем выше температура, при которой последовательность специфически гибридизуется с ее целевой последовательностью. Неограничивающие примеры жестких условий подробно описаны в Tijssen (1993), Laboratory Techniques In Biochemistry And Molecular Biology-Hybridization With Nucleic Acid Probes Part I, Second Chapter "Overview of principles of hybridization and the strategy of nucleic acid probe assay", Elsevier, N.Y. Когда приводится полинуклеотидная последовательность, то также предусматриваются комплементарные или частично комплементарные последовательности. Они предпочтительно способны гибридизироваться с упоминаемой последовательностью в очень жестких условиях. Как правило, для максимизации скорости гибридизации выбирают условия относительно низкой гибридизации: примерно на 20-25°C ниже, чем термическая температура плавления (Tm). Tm представляет собой температуру, при которой 50% конкретной последовательности-мишени гибридизуется с идеально комплементарным зондом в растворе при определенной ионной силе и рН. Как правило, для того, чтобы требовать по меньшей мере ~85% нуклеотидной комплементарности гибридизованных последовательностей, выбирают очень жесткие условия промывки примерно на 5-15°C ниже, чем Tm. Чтобы требовать по меньшей мере приблизительно 70% нуклеотидной комплементарности гибридизованных последовательностей, выбирают умеренно жесткие условия промывки примерно на 15-30°C ниже, чем Tm. Условия высокой разрешимости (очень низкая жесткость) могут быть на 50°C ниже Tm, что обеспечивает высокий уровень ошибочного соответствия между гибридизованными последовательностями. Специалисты в данной области понимают, что другие физические и химические параметры на этапах гибридизации и промывки также могут быть изменены для влияния на результат детектируемого сигнала гибридизации с определенного уровня гомологии между последовательностями-мишенями и зондами. Предпочтительные очень жесткие условия включают инкубацию в 50% формамиде, 5×SSC и 1% SDS при 42°C или инкубацию в 5×SSC и 1% SDS при 65°C с промывкой 0,2×SSC и 0,1% SDS при 65°C. "Гибридизация" относится к реакции, в которой один или более полинуклеотидов реагируют с образованием комплекса, который стабилизируется посредством водородной связи между основаниями нуклеотидных остатков. Водородное связывание может происходить по принципу спаривания оснований Уотсона-Крика, хугстиновского связывания или любым другим способом, специфичным для последовательности. Комплекс может содержать две цепи, образующие дуплексную структуру, три или более цепей, образующих многоцепочечный комплекс, одну цепь, которая гибридизируется сама с собой, или любую их комбинацию. Реакция гибридизации может представлять собой стадию в более широком процессе, таком как инициирование ПЦР или расщепление полинуклеотида ферментом. Последовательность, способная к гибридизации с данной последовательностью, называется "комплементарной" данной последовательности. Используемый в настоящем описании термин "геномный локус" или "локус" (множественное число: локусы) представляет собой специфическое местоположение гена или последовательности ДНК на хромосоме. "Ген" относится к участкам ДНК или РНК, которые кодируют полипептид или цепь РНК, которая играет функциональную роль в организме и, следовательно, является молекулярной единицей наследственности в живых организмах. Для целей настоящего изобретения можно считать, что гены включают области, которые регулируют продукцию гена, независимо от того, являются ли такие регулятивные последовательности "смежными" с кодирующими и/или транскрибируемыми последовательностями. Соответственно, ген включает, но не обязательно ограничивается ими, последовательности промоторов, терминаторы, последовательносты регуляторов трансляции, такими как сайты связывания рибосом и внутренние сайты входа рибосомы, энхансеры, сайленсеры, изоляторы, пограничные элементы, точки начала репликации, сайты прикрепления матрикса и области контроля локуса. Как используют в настоящем описании, термин "экспрессия геномного локуса" или "экспрессия гена" представляет собой процесс, посредством которого информация, заключенная в гене, используется для синтеза функционального генного продукта. Продукты экспрессии генов часто являются белками, но в генах, не содержащих белок, таких как гены рРНК или гены тРНК, продукт представляет собой функциональную РНК. Процесс экспрессии генов используется всеми известными живыми организмами - эукариотами (включая многоклеточные организмы), прокариотами (бактериями и археями) и вирусами для выработки функциональных продуктов для выживания. Как используют в настоящем описании, термин "экспрессия" гена или нуклеиновой кислоты охватывает не только экспрессию клеточного гена, но также транскрипцию и трансляцию нуклеиновой кислоты (кислот) в системах клонирования и в любом другом контексте. Как используют в настоящем описании, термин "экспрессия" также относится к способу, посредством которого полинуклеотид транскрибируется из ДНК-матрицы (например, в мРНК или другой транскрипт РНК), и/или к процессу, посредством которого транскрибированная мРНК затем транслируется в пептиды, полипептиды или белки. Транскрипты и кодированные полипептиды могут в совокупности обозначаться как "генный продукт". Если полинуклеотид получен из геномной ДНК, экспрессия может включать сплайсинг мРНК в эукариотической клетке. Термины "полипептид", "пептид" и "белок" используют в настоящем описании взаимозаменяемо для обозначения полимеров аминокислот любой длины. Полимер может быть линейным или разветвленным, он может содержать модифицированные аминокислоты и может быть прерван неаминокислотами. Термины также включают модифицированный аминокислотный полимер; например, образование дисульфидной связи, гликозилирование, липидацию, ацетилирование, фосфорилирование или любые другие манипуляции, такие как конъюгация с меченым компонентом. Как используют в настоящем описании, термин "аминокислота" включает природные и/или неприродные или синтетические аминокислоты, включая глицин и оба оптических изомера D или L, а также аминокислотные аналоги и пептидомиметики. Как используют в настоящем описании, термин "домен" или "белковый домен" относится к части белковой последовательности, которая может существовать и функционировать независимо от остальной белковой цепи. Как описано для аспектов данного изобретения, идентичность последовательности связана с гомологией последовательности. Сравнения гомологии могут проводиться на глаз или, чаще всего, с помощью легкодоступных программ сравнения последовательностей. Эти коммерчески доступные компьютерные программы могут вычислять процентную (%) гомологию между двумя или более последовательностями и также могут вычислять идентичность последовательностей, которой обладают две или более последовательностей аминокислот или нуклеиновых кислот.
[670] В определенных вариантах изобретения термин "направляющая РНК" относится к полинуклеотидной последовательности, содержащей одну или более предполагаемых или идентифицированных tracr-последовательностей и предполагаемых или идентифицированных последовательностей cr-РНК или направляющей последовательности. В конкретных вариантах осуществления "направляющая РНК" включает предполагаемую или идентифицированную последовательность cr-РНК или направляющую последовательность. В других вариантах осуществления направляющая РНК не включает предполагаемую или идентифицированную tracr-последовательность.
[671] Как используют в настоящем описании, термин "дикий тип" является термином, понятным квалифицированным специалистам, и означает типичную форму организма, штамма, гена или характеристики в том виде, как они встречаются в природе, в отличие от мутантных или вариантных форм. "Дикий тип" может быть базовой линией.
[672] Как используют в настоящем описании, термин "вариант" следует понимать как проявление свойств, которые имеют характерные черты, по которым эти свойства отличаются от природных свойств.
[673] Термины "неприродный" или "сконструированный способами инженерии" используются взаимозаменяемо и указывают на вмешательство человека. Эти термины, относящиеся к молекулам нуклеиновой кислоты или полипептидам, означают, что молекула нуклеиновой кислоты или полипептид по меньшей мере в некоторой степени лишены одного другого компонента, с которым они естественным образом ассоциированы в своем природном виде. Во всех аспектах и вариантах осуществления, независимо от того, включают ли они эти термины или нет, следует понимать, что использование этих терминов необязательно и, следовательно, не существует предпочтения по их включению. Кроме того, термины "неприродный" или "полученный способами инженерии" могут использоваться взаимозаменяемо и поэтому могут применяться по отдельности или вместе, и то или другое может заменить упоминание обоих вместе. В частности, "полученный способами инженерии" предпочтительнее вместо "неприродный" или "неприродный и/или полученный способами инженерии".
[674] Гомология последовательностей может быть определена любой из ряда компьютерных программ, известных в данной области, например BLAST или FASTA et al. Подходящей для выполнения такого выравнивания компьютерной программой является пакет GCG Wisconsin Bestfit (Университет Висконсина, США, University of Wisconsin, U.S.A; Devereux et al., 1984, Nucleic Acids Research 12:387). Примеры другого программного обеспечения, способного выполнять сравнения последовательностей, включают, без наложения ограничений, пакет BLAST (см. Ausubel et al., 1999 ibid - Chapter 18), FASTA (Atschul et al., 1990, J. Mol. Biol., 403-410) и набор инструментов сравнения GENEWORKS. Как BLAST, так и FASTA доступны для поиска оффлайн и онлайн (см. Ausubel et al., 1999 ibid, pages 7-58 to 7-60). При этом предпочтительно использование программы GCG Bestfit. Процент (%) гомологии последовательно может быть рассчитан для перекрывающихся последовательностей, т.е. одна последовательность выровнена против другой и аминокислота или нуклеотид одной последовательности напрямую сравнивается с соответствующей другой аминокислотой или нуклеотидом другой последовательности по одной(му) за раз. В этом случае его называют "выравнивание без пропусков". Обычно такое выравнивание без пропусков выполнятся только для сравнительно небольшого числа остатков. Хотя это очень простой и логичный способ, он не способен рассматривать, к примеру, случаи идентичных в других отношениях пары последовательностей, где одна инсерция или делеция может привести к тому, что последующий аминокислотный остаток окажется вне рассмотрения выравнивания, тем самым приводя к возможному большому уменьшению % гомологии при выполнении глобального выравнивания. В результате большинство способов сравнения последовательностей разработаны для проведения оптимального выравнивания с учетом возможных инсерций и делеций без необоснованного наложения штрафа общей гомологии или балла идентичности. Это достигается путем вставления "пропусков" в выравнивании последовательности с целью максимизации локальной гомологии или идентичности. Однако такие более сложные способы налагают "штраф за внесение пропуска" для каждого пропуска, возникающего в выравнивании, так что для того же числа идентичных аминокислот возможно добавление нескольких пропусков - что отражает более близкое родство между двумя сравниваемыми последовательностями - и позволяет достичь более высокого балла, чем в случае одной со многими пропусками. "Цена сродства пропусков" обычно используется для наложения сравнительно большого штрафа за присутствие пропуска и меньшее - за каждый последующий остаток в пропуске. Это наиболее часто используемая система оценки пропусков. Высокие штрафы за пропуски могут, разумеется, давать в результате оптимизированные выравнивания с меньшим числом пропусков. Большинство программ выравнивания допускают внесение изменений в штрафы за пропуски. Однако предпочтительно использование исходных значений при использовании такого программного обеспечения для сравнения последовательностей. Например, при использовании пакета GCG Wisconsin Bestfit исходный штраф за пропуск в аминокислотных последовательностях равен -12 за каждый пропуск и -4 за каждую вставку. Расчет максимального % гомологии, таким образом, сначала требует проведения оптимального выравнивания с рассмотрением штрафов за пропуски. Подходящая компьютерная программа для проведения такого выравнивая - пакет GCG Wisconsin Bestfit (Devereux et al., 1984 Niic. Acids Research 12 p387). Примерами другого программного обеспечения, способного выполнять сравнение последовательностей являются, но не ограничиваются ими, пакеты BLAST (см. Ausubel et al., 1999 Short Protocols in Molecular Biology, 4to Ed. - Chapter 18), FASTA (Altschul et al., 1990 J. Mol Biol. 403-410) и набор инструментов сравнения GENEWORKS. Как BLAST, так и FASTA доступны для поиска оффлайн и онлайн (см. Ausubel et al., 1999 ibid, pages 7-58 to 7-60). Однако для решения некоторых задач предпочтительно использование программы GCG Bestfit. Новый инструмент, называющийся BLAST 2 Sequences, также используется для сравнения белковых и нуклеотидных последовательностей (см. FEMS Microbiol Lett. 1999 174(2): 247-50; FEMS Microbiol Lett. 1999 177(1): 187-8 и веб-сайт Национального Центра Биотехнологии на веб-сайте Национального Института Здравоохранения США). Хотя конечный % гомологии может быть измерен в смысле идентичности, процесс проведения выравнивания сам по себе не основан на сравнении "все-или-ничего" пар. Вместо этого обычно используется масштабированная матрица оценки сходства для расчета баллов для каждого попарного сравнения на основе химического сходства или эволюционного расстояния. Примером такой часто используемой матрицы является матрица BLOSUM62 - матрица, используемая по умолчанию для набора программ BLAST. Программы GCG Wisconsin обычно используют либо общедоступные значения по умолчанию, либо пользовательскую таблицу сравнения символов при ее наличии (см. руководство пользователя для дальнейших деталей). Для решения некоторых задач предпочтительно использование общедоступных значений по умолчанию пакета GCG или, в случае другого программного обеспечения, матрицы по умолчанию, такой как BLOSUM62. Альтернативно этому, процент гомологии может быть рассчитан с использованием свойства множественного выравнивания из DNASIS™ (Hitachi Software), основанного на алгоритме, аналогичном CLUSTAL (Higgins DG & Sharp PIVI (1988), Gene 73(1), 237-244). После того как программное обеспечение нашло оптимальное выравнивание, становится возможным рассчитать % гомологии, предпочтительно % идентичности последовательности. Данное программное обеспечение обычно производит это в ходе сравнения последовательностей и приводит численный результат. Последовательности могут также иметь делеции, инсерции или замены аминокислотных остатков, что приводит к изменению без уведомления и получению в результате функционально эквивалентных состояний. Преднамеренные замены аминокислот могут быть сделаны на основе сходства в свойствах аминокислот (таких как полярность, заряд, растворимость, гидрофобность, гидрофильность и/или амфипатическая природа остатков) и, как следствие, полезны для объединения аминокислот в функциональные группы. Аминокислоты могут быть сгруппированы на основе свойств только их боковых цепей. Однако более целесообразно также включать данные о мутациях. Наборы аминокислот, полученные таким образом, с большой вероятностью являются консервативными в связи с их структурой. Такие наборы могут быть описаны в форме диаграмм Венна (Livingstone C.D. and Barton G.J. (1993) "Protein sequence alignments: a strategy for the hierarchical analysis of residue conservation" Comput. Appl. Biosci. 9: 745-756) (Taylor W.R. (1986) "The classification of amino acid conservation" J. Theor. Biol 119, 205-218). Могут быть произведены консервативные замены, например в соответствии с таблицей ниже, которая описывает общепринятую группировку аминокислот в виде диаграммы Венна.
[675] Термины "субъект", "индивид" и "пациент" используются в настоящем описании взаимозаменяемо для обозначения позвоночного, предпочтительно млекопитающего, более предпочтительно человека. Млекопитающие включают, но не ограничиваются ими, мышей, обезьян, людей, сельскохозяйственных животных, спортивных животных и домашних животных. Также охватываются ткани, клетки и их потомки, полученные от биологического объекта in vivo или культивируемые in vitro.
[676] Термины "терапевтический агент", "терапевтически способный агент" или "лечебный агент" используются взаимозаменяемо и относятся к молекуле или соединению, которые придают какой-либо полезный эффект при введении индивиду. Полезный эффект включает обеспечение диагностических определений; облегчение протекания заболевания, симптома, расстройства или патологического состояния; уменьшение или предотвращение возникновения заболевания, симптома, расстройства или состояния; и в целом противодействие заболеванию, симптому, расстройству или патологическому состоянию.
[677] Как используют в настоящем описании, термины "лечение" или "облегчение" или "улучшение" используются взаимозаменяемо. Эти термины относятся к подходу для получения полезных или желаемых результатов, включая, но не ограничиваясь ими, терапевтический эффект и/или профилактическую пользу. Под терапевтическим эффектом понимается любое терапевтически значимое улучшение или воздействие на одно или более заболеваний, состояний или симптомов, подлежащих лечению. Для профилактической пользы конструкции могут вводиться индивиду, подверженному риску развития конкретного заболевания, состояния или симптома, или индивиду, демонстрирующему один или более физиологических симптомов заболевания, хотя болезнь, состояние или симптом, возможно, еще не проявились.
[678] Термин "эффективное количество" или "терапевтически эффективное количество" относится к количеству агента, достаточному для получения полезных или желаемых результатов. Терапевтически эффективное количество может варьироваться в зависимости от одного или более факторов: индивид и состояние заболевания, подвергаемого лечению, вес и возраст индивида, тяжесть заболевания, способ введения и т.п., которые могут быть легко определены специалистом в данной области. Этот термин также относится к дозе, которая обеспечит визуализацию для обнаружения любым из способов визуализации, описанных в настоящем описании. Конкретная доза может варьироваться в зависимости от одного или более факторов: выбранный конкретный агент, режим дозирования, который следует соблюдать, введение комбинации с другими соединениями, время введения, подлежащая визуализации ткань и физическая система доставки.
[679] В практике настоящего изобретения используются, если не указано иначе, общепринятые способы иммунологии, биохимии, химии, молекулярной биологии, микробиологии, клеточной биологии, геномики и рекомбинантных ДНК, которые входят в компетенции специалистов. См. Sambrook, Fritsch and Maniatis, MOLECULAR CLONING: A LABORATORY MANUAL, 2nd edition (1989); CURRENT PROTOCOLS IN MOLECULAR BIOLOGY (F, M. Ausubel, et al. eds., (1987)); the series METHODS IN ENZYMOLOGY (Academic Press, Inc.): PCR 2: A PRACTICAL APPROACH (M.J. MacPherson, B.D. Flames and G.R. Taylor eds. (1995)), Harlow and Lane, eds. (1988) ANTIBODIES, A LABORATORY MANUAL, and ANIMAL CELL CULTURE (R.I. Freshney, ed. (1987)).
[680] Несколько аспектов изобретения относятся к векторным системам, содержащим один или более векторов, или к векторам как таковым. Векторы могут быть разработаны для экспрессии транскриптов CRISPR (например, транскриптов нуклеиновых кислот, белков или ферментов) в прокариотических или эукариотических клетках. Например, транскрипты CRISPR могут быть экспрессированы в бактериальных клетках, таких как Escherichia coli, клетках насекомых (с использованием экспрессирующих векторов бакуловируса), дрожжевых клетках или клетках млекопитающих. Подходящие клетки-хозяева обсуждаются далее в Goeddel, GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990). Альтернативно, рекомбинантный экспрессирующий вектор можно транскрибировать и транслировать in vitro, например, с использованием регуляторных последовательностей промотора T7 и полимеразы T7.
[681] Варианты осуществления изобретения включают последовательности (как полинуклеотидные, так и полипептидные), которые могут содержать гомологичную замену ("замещение" используются в настоящем описании для обозначения обмена существующего аминокислотного остатка или нуклеотида с альтернативным остатком или нуклеотидом), которая может происходить, например, для подобной замены в случае аминокислот, таких как основные для основных, кислотные для кислотных, полярные для полярных и т.д. Негомологичное замещение может также происходить, то есть из одного класса остатков в другой или, в качестве альтернативы, включение неестественных аминокислот, таких как орнитин (далее обозначаемый как Z), орнитин диаминомасляной кислоты (далее называемый B), норлейцин орнитин (далее называемый O), пироариланин, тиенилаланин, нафтилаланин и фенилглицин. Вариантные аминокислотные последовательности могут включать подходящие спейсерные группы, которые могут быть вставлены между любыми двумя аминокислотными остатками последовательности, включая алкильные группы, такие как метил-, этил- или пропильные группы, в дополнение к аминокислотным спейсерам, таким как остатки глицина или β-аланина. Специалисты в данной области могут хорошо понимать дальнейшую форму вариации, которая включает присутствие одного или более аминокислотных остатков в пептоидной форме. Во избежание сомнений, "пептоидная форма" используется для обозначения вариантов аминокислотных остатков, в которых α-углеродная заместительная группа находится на атоме азота остатка, а не на α-углероде. Способы получения пептидов в пептоидной форме известны в данной области, например, Simon RJ et al., PNAS (1992) 89 (20), 9367-9371 и Horwell DC, Trends Biotechnol. (1995) 13 (4), 132-134.
[682] Моделирование гомологии: соответствующие остатки в других ортологах C2c2 могут быть идентифицированы с помощью способов Zhang et al., 2012 (Nature, 490 (7421): 556-60) и Chen et al., 2015 (PLoS Comput Biol; 11 (5): e1004248) - способ вычисления белок-белкового взаимодействия (PP1) для прогнозирования взаимодействий, опосредованных поверхностями контакта домен-мотив. PrePPI (Predicting PPI), основанный на структуре способ прогнозирования PPI, объединяет структурные доказательства с неструктурными данными с использованием байесовской статистической модели. Способ включает взятие пары белков запроса и использование структурного выравнивания для идентификации структурных представителей, соответствующим либо их экспериментально определенным структурам, либо гомологичным моделям. Структурное выравнивание далее используется для идентификации как близких, так и отдаленных структурных соседей путем рассмотрения глобальных и локальных геометрических отношений. Всякий раз, когда два соседа структурных представителей образуют комплекс, указанный в Protein Data Bank, это определяет шаблон для моделирования взаимодействия между двумя белками запросов. Модели комплекса создаются путем наложения репрезентативных структур на соответствующего структурного соседа в шаблоне. Этот подход далее описан в Dey et al, 2013 (Prot Sci, 22: 359-66).
[683] Для целей настоящего изобретения амплификация означает любой способ, использующий праймер и полимеразу, способную реплицировать последовательность-мишень с разумной точностью. Амплификацию можно проводить с помощью природных или рекомбинантных ДНК-полимераз, таких как ДНК-полимераза TaqGold™, T7, фрагмент Кленова ДНК-полимеразы E.coli и обратная транскриптаза. Предпочтительным способом амплификации является ПЦР.
[684] В некоторых вариантах изобретение включает векторы. Как используют в настоящем описании, термин "вектор" представляет собой инструмент, который позволяет или облегчает передачу объекта из одной среды в другую. Это репликон, такой как плазмида, фаг или космида, в которые может быть вставлен другой сегмент ДНК, чтобы вызвать репликацию вставленного сегмента. Как правило, вектор способен к репликации, когда он связан с соответствующими элементами управления. В общем, термин "вектор" относится к молекуле нуклеиновой кислоты, способной транспортировать другую нуклеиновую кислоту, с которой она была связана. Векторы включают, но не ограничиваются, молекулами нуклеиновых кислот, которые являются однонитевыми, двухцепочечными или частично двухцепочечными; молекулами нуклеиновых кислот, которые содержат один или более свободных концов, или не содержат свободных концов (например, круглые); молекулы нуклеиновых кислот, которые содержат ДНК, РНК или и то, и другое; и другие разновидности полинуклеотидов, известных в данной области. Один тип вектора представляет собой "плазмиду", которая относится к круглой двухцепочечной петле ДНК, в которую могут быть добавлены дополнительные сегменты ДНК стандартными способами молекулярного клонирования. Другим типом вектора является вирусный вектор, в котором в векторе для упаковки в вирус присутствуют вирусные ДНК или РНК (например, ретровирусы, ретровирусы с дефектами репликации, аденовирусы, аденовирусы с дефектами репликации и аденоассоциированные вирусы (AAV)). Вирусные векторы также включают полинуклеотиды, переносимые вирусом для трансфекции в клетку-хозяина. Некоторые векторы способны к автономной репликации в клетке-хозяине, в которую они вводятся (например, бактериальные векторы, имеющие бактериальную точку начала репликации и эписомальные векторы млекопитающих). Другие векторы (например, неэписомальные векторы млекопитающих) интегрируются в геном клетки-хозяина, после введения в клетку-хозяина и, таким образом, реплицируются вместе с геномом хозяина. Более того, некоторые векторы способны направлять экспрессию генов, с которыми они функционально связаны. Такие векторы упоминаются в настоящем описании как "экспрессирующие векторы". Обычные экспрессирующие векторы, использующиеся в способах рекомбинантной ДНК, часто имеют форму плазмид.
[685] Рекомбинантные экспрессирующие векторы могут содержать нуклеиновые кислоты по изобретению в форме, подходящей для экспрессии нуклеиновых кислот в клетке-хозяине, что означает, что рекомбинантные экспрессирующие векторы включают один или более регуляторных элементов, которые могут быть выбраны на основе клеток-хозяев, используемых для экспрессии, и которые функционально связаны с последовательностью нуклеиновых кислот, которая должна быть экспрессирована. Внутри рекомбинантного экспрессирующего вектора "функционально связанный" означает, что целевая нуклеотидная последовательность связана с регуляторным элементом(ами) таким образом, который позволяет экспрессию нуклеотидной последовательности (например, в системе in vitro транскрипции/трансляции или в клетке-хозяине, когда вектор вводится в клетку-хозяина). Что касается способов рекомбинации и клонирования, может быть упомянута заявка на патент США 10/815730, опубликованная 2 сентября 2004 года в качестве US 2004-0171156 A1, содержание которой включено в настоящее описание в качестве ссылки в полом объеме.
[686] Аспекты изобретения относятся к бицистронным векторам для направляющей РНК и РНК дикого типа, модифицированных или мутированных эффекторных белков/ферментов CRISPR (например, C2c2). Бицистронные экспрессирующие векторы направляющей РНК и РНК дикого типа, модифицированных или мутированных эффекторных белков/ферментов CRISPR (например, C2c2). В целом и, в частности, в этом варианте осуществления и диком типе, модифицированные или мутированные эффекторные белки/ферменты CRISPR (например, C2c2) предпочтительно находятся под контролем промотора CBh. РНК может предпочтительно находиться под контролем промотора РНК-полимеразы III, такого как промотор U6. В идеале эти два промотора сочетаются.
[687] В некоторых вариантах осуществления предусмотрена петля в направляющей РНК. Это может быть шпилька или тетрапетля. Петля предпочтительно представляет собой GAAA, но не ограничивается этой последовательностью или даже длиной 4 п.о. Действительно, предпочтительные последовательности формирования петли для использования в структурах шпильки имеют длину четыре нуклеотида и наиболее предпочтительно имеют последовательность GAAA. Однако могут использоваться более длинные или более короткие последовательности петель, а также альтернативные последовательности. Последовательности предпочтительно включают нуклеотидный триплет (например, AAA) и дополнительный нуклеотид (например, C или G). Примеры последовательностей, формирующих петли, включают CAAA и AAAG.
[688] При осуществления любого из способов, описанных в настоящем описании, подходящий вектор может быть введен в клетку или эмбрион посредством одного или более способов, известных в данной области, включая, но не ограничиваясь ими, микроинъекцию, электропорацию, сонопорацию, биолистику, опосредованную фосфатом кальция трансфекцию, катионную трансфекцию, трансфекцию липосом, трансфекцию дендримера, трансфекцию теплового шока, нуклеофекцию, магнитотрансфекцию, липофекцию, импалефекцию, оптическую трансфекцию, запатентованное усиленное агентом поглощение нуклеиновых кислот, доставку посредством липосом, иммунолипосом, виросом или искусственных вирионов. В некоторых способах вектор вводится в эмбрион путем микроинъекции. Вектор или векторы могут быть введены посредством микроинъекции в ядро или цитоплазму эмбриона. В некоторых способах вектор или векторы могут быть введены в клетку путем нуклеофекции.
[689] Термин "регуляторный элемент" используется для обозначения промоторов, энхансеров, участков внутренней посадки рибосомы (IRES) и других элементов управления экспрессией (например, сигналов терминации транскрипции, таких как сигналы полиаденилирования и последовательности поли-U). Такие регуляторные элементы описаны, например, в Goeddel, GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, San Diego, Calif. (1990). Регуляторы включают как те, которые напрямую определяют конститутивную экспрессию нуклеотидной последовательности во многих типах клеток-хозяев, так и те, которые управляют экспрессией нуклеотидной последовательности только в определенных клетках-хозяевах (например, тканеспецифические регуляторные последовательности). Тканеспецифический промотор может управлять экспрессией в основном в желаемой интересующей ткани, такой как мышца, нейрон, кость, кожа, кровь, специфические органы (например, печень, поджелудочная железа) или конкретные типы клеток (например, лимфоциты). Регуляторные элементы также могут управлять экспрессией в зависимости от времени, например, в зависимости от стадии клеточного цикла или стадии развития, и могут быть или не быть специфичным для тканей или клеток. В некоторых вариантах осуществления вектор содержит один или более промоторов ДНК-полимеразы III (например, 1, 2, 3, 4, 5 или более промоторов ДНК-полимеразы III), один или более промоторов ДНК-полимеразы II (например, 1, 2, 3, 4, 5 или более промоторов ДНК-полимеразы II), один или более промоторов ДНК-полимеразы I (например, 1, 2, 3, 4, 5 или более промоторов ДНК-полимеразы I) или их комбинации. Примеры промоторов ДНК-полимеразы III включают, но не ограничиваются ими, промоторы U6 и HI. Примеры промоторов ДНК-полимеразы II включают, но не ограничиваются ими, ретровирусный промотор LTR RAR-саркомы Рауса (RSV) (необязательно с энхансером RSV), промотор цитомегаловируса (CMV) (необязательно с энхансером CMV) [см., например, Boshart et al., Ceil, 41: 521-530 (1985)], промотор SV40, промотор дигидрофолатредуктазы, промотор 3-актина, промотор фосфоглицерин-киназы (PGK) и промотор EF1a. Кроме того, термин "регуляторный элемент" охватывает энхансерные элементы, такие как WPRE; энхансеры CMV; сегмент R-U5' в LTR HTLV-I (Mol, Cell. Biol, том 8 (1), стр. 466-472, 1988); энхансер SV40; и интронная последовательность между экзонами 2 и 3 белка кролика P-глобина (Proc. Natl. Acad. Sci. USA., т. 78 (3), p, 1527-31, 1981). Специалистам в данной области будет понятно, что конструкция вектора экспрессии может зависеть от таких факторов, как выбор клетки-хозяина, которая должна быть трансфицирована, а также желаемый уровень экспрессии и т.д. Вектор может быть введен в клетки таким образом, чтобы они продуцировали транскрипты, белки или пептиды, включая слитые белки или пептиды, кодируемые нуклеиновыми кислотами, как описано в настоящем описании (например, транскрипты системы коротких палиндромных повторов, регулярно расположенных кластерами (CRISPR), их белки, ферменты, их мутантные формы, их слитые белки и т.д.). Что касается регуляторных последовательностей, то может быть упомянута заявка на патент США 10/491026, содержание которой включено в настоящее описание в качестве ссылки в полном объеме. Что касается промоторов, упоминается публикация РСТ WO 2011/028929 и Приложение США 12/511940, содержание которых включено в настоящее в качестве ссылки в полном объеме.
[690] Векторы могут быть сконструированы для экспрессии транскриптов CRISPR (например, транскриптов нуклеиновых кислот, белков или ферментов) в прокариотических или эукариотических клетках. Например, транскрипты CRISPR могут быть экспрессированы в бактериальных клетках, таких как Escherichia coli, клетках насекомых (с использованием экспрессирующих векторов бакуловируса), дрожжевых клетках или клетках млекопитающих. Подходящие клетки обсуждаются далее в Goeddel, GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY185, Academic Press, San Diego, Calif. (1990). Альтернативно, рекомбинантный экспрессирующий вектор можно транскрибировать и транслировать in vitro, например, с использованием регуляторных последовательностей промотора T7 и полимеразы T7.
[691] Векторы могут введены и увеличены в количестве в прокариотических организмах или прокариотических клетках. В некоторых вариантах осуществления прокариотический организм используют для амплификации копий вектора, который должен быть введен в эукариотическую клетку или в качестве промежуточного вектора при образовании вектора, который должен быть введен в эукариотическую клетку (например, амплификация плазмиды как части вирусной векторная упаковочная система). В некоторых вариантах осуществления прокариотический организм используется для амплификации копий вектора и экспрессии одной или более нуклеиновых кислот, например, для обеспечения источника одного или более белков для доставки в клетку-хозяина или организм-хозяин. Экспрессия белков в прокариотах чаще всего происходит в Escherichia coli с векторами, содержащими конститутивные или индуцируемые промоторы, которые направляют экспрессию либо слитых, либо неслитых белков. Слитые векторы встраивают ряд аминокислот в кодируемый им белок, например, на N-конце рекомбинантного белка. Такие слитые векторы могут служить одной или более целям, таким как: (i) увеличение экспрессии рекомбинантного белка; (ii) увеличение растворимости рекомбинантного белка; и (iii) содействие очистке рекомбинантного белка, действуя как лиганд в аффинной очистке. Часто в слитых экспрессирующих векторах вводится сайт протеолитического расщепления на стыке области слияния и рекомбинантного белка, чтобы обеспечить отделение рекомбинантного белка от слитого фрагмента после очистки слитого белка. Такие ферменты и их родственные последовательности распознавания включают фактор Ха, тромбин и энтерокиназу. Примеры экспрессирующих векторов слияния включают pGEX (Pharmacia Biotech Inc, Smith and Johnson, 1988. Gene 67: 31-40), pMAL (New England Biolabs, Beverly, Mass.) И pRIT5 (Pharmacia, Piscataway, NJ), которые производят слияние глютатион S-трансферазы (GST), связывающего мальтозу белка E или белка A, соответственно, с целевым рекомбинантным белком.
[692] Примеры подходящих индуцибельных неслитых экспрессирующих векторов E.coli включают pTrc (Amrann et al., (1988) Gene 69: 301-315) и pET lid (Studier et al., GENE EXPRESSION TECHNOLOGY: METHODS IN ENZYMOLOGY 185, Academic Press, Сан-Диего, Калифорния (1990) 60-89).
[693] В некоторых вариантах осуществления вектор представляет собой экспрессирующий вектор дрожжей. Примеры экспрессирующих векторов в дрожжах Saccharomyces cerivisae включают pYepSeel (Baldari, et al., 1987. EMBO J. 6: 229-234), pMFa (Kuijan and Herskowitz, 1982. Cell 30: 933-943), pJRY88 (Schultz et al., 1987. Gene 54: 113-123), pYES2 (Invitrogen Corporation, San Diego, CA.) и picZ (InVitrogen Corp, Сан-Диего, Калифорния).
[694] В некоторых вариантах осуществления вектор управляет экспрессией белка в клетках насекомых с использованием экспрессирующих векторов бакуловируса. Бакуловирусные векторы, доступные для экспрессии белков в культивируемых клетках насекомых (например, клетки SF9), включают серию pAc (Smith, et al., 1983. Mol. Cell. Biol., 3: 2156-2165) и серию pVL (Lucklow and Summers, 1989. Virology 170: 31-39).
[695] В некоторых вариантах осуществления вектор способен приводить к экспрессии одной или более последовательностей в клетках млекопитающих с использованием вектора экспрессии в млекопитающих. Примеры экспрессирующих векторов млекопитающих включают pCDM8 (Seed, 1987. Nature 329: 840) и pMT2PC (Kaufman, et al., 1987. EMBO J. 6: 187-195). При использовании в клетках млекопитающих функции управления вектором экспрессии обычно выполняются одним или более регуляторными элементами. Например, обычно используемые промоторы получают из полиомы, аденовируса 2, цитомегаловируса, вируса SV40 и других, описанных в настоящем описании и известных в данной области. Другие подходящие системы экспрессии как для прокариотических, так и для эукариотических клеток можно изучить по: см., например, главы 16 и 17 Sambrook et al. MOLECULAR CLONING: A LABORATORY MANUAL, 2nd ed., Laboratory Cold Spring Harbor, Laboratory Cold Spring Harbour Press, Cold Spring Harbour, NY, 1989.
[696] В некоторых вариантах осуществления рекомбинантный экспрессирующий вектор млекопитающих способен направлять экспрессию нуклеиновых кислот предпочтительно в конкретном типе клеток (например, тканеспецифические регуляторные элементы используются для экспрессии нуклеиновой кислоты). Специфические для ткани регуляторные элементы известны в данной области. Не ограничивающие примеры подходящих тканеспецифических промоторов включают промотор альбумина (специфический для печени: Pinkert, et al., 1987. Genes Dev. 1: 268-277), лимфоидспецифические промоторы (Calarne and Eaton, 1988. Adv. Immunol 43: 235-275), в частности, промоторы рецепторов Т-клеток (Winoto and Baltimore, 1989. EMBO J. 8: 729-733) и иммуноглобулины (Baneiji, et al., 1983. Cell 33: 729-740; Queen and Baltimore, 1983. Cell 33: 741-748), нейроспецифические промоторы (например, промотор нейрофиламентов, Byrne and Ruddle, 1989. Proc. Natl. Acad. Sci. USA, 86: 5473-5477), специфические для поджелудочной железы промоторы (Edlund, et al., 1985. Science 230: 912-916) и промоторы молочной железы, специфические для молочной железы (например, промотор молочной сыворотки, патент США №4873,316 и публикация Европейской заявки №264,166). Регулируемые развитием организма промоторы также включают, например, hox-промоторы мыши (Kessel and Grass, 1990. Science 249: 374-379) и промотор α-фетопротеина (Campes and Tilghman, 1989. Гены Dev. 3: 537-546). Что касается этих прокариотических и эукариотических векторов, то может быть упомянут патент США 6750509, содержание которого включено в настоящее описание в качестве ссылки в полном объеме полноте. Другие варианты осуществления изобретения могут относиться к использованию вирусных векторов, в отношении которых упоминается заявка на патент США 13/1002085, содержание которой включено в настоящее описание посредством ссылки в полном объеме. Специфичные для ткани регуляторные элементы известны в данной области, и в этой связи упоминается патент США 7776321, содержание которого включено в настоящее описание посредством ссылки в полном объеме.
[697] В некоторых вариантах осуществления регуляторный элемент функционально связан с одним или более элементами системы CRISPR, чтобы управлять экспрессией одного или более элементов системы CRISPR. В целом, CRISPR (короткие палиндромные повторы, регулярно расположенные кластерами), также известный как SPIDR (рассеянные между спейсерами прямые повторы), представляют собой семейство локусов ДНК, которые обычно специфичны для конкретных видов бактерий. Локус CRISPR содержит отчетливый класс чередующихся коротких повторов последовательности (SSR), которые были описаны в E. coli (Ishino et al., J. Bacteriol. 169: 5429-5433 [1987] и Nakata et al., J. Bacteriol., 171: 3553-3556 [1989]) и ассоциированных генов. Аналогичные участки SSR были идентифицированы в Haloferax mediterranei, Streptococcus pyogenes, Anabaena и Mycobacterium tuberculosis (см. Groenen et al., Mol. Microbiol., 10: 1057-1065 [1993], Hoe et al., Emerg. Infect. Dis., 5: 254-263 [1999], Masepohl et al., Biochim Biophys Acta 1307: 26-30 [1996], Mojica et al., Mol. Microbiol., 17: 85-93 [1995]). Локусы CRISPR, как правило, отличаются от других SSR структурой повторов, которые называются короткими регулярно прерывающимися повторами (SRSR) (Janssen et al., OMICS J. Integ. Biol., 6: 23-33 [2002], и Mojica et al., Mol. Microbiol., 36: 244-246 [2000]). В общем случае, повторы представляют собой короткие элементы, которые образуют кластеры, которые регулярно распределены по уникальным перемежаемым последовательностям с примерно постоянной длиной (Mojica et al., [2000], см. выше). Хотя повторяющиеся последовательности очень консервативны для разных штаммов, количество прерывающихся повторов и последовательностей спейсерных областей обычно отличаются для разных штаммов (van Embden et al., J. Bacteriol, 182: 2393-2401 [2000]). Локусы CRISPR были идентифицированы более чем у 40 прокариотов (см., Например, Jansen et al., Mol. Microbiol., 43: 1565-1575 [2002] и Mojica et al., [2005]), включая, но не ограничиваясь, Aeropyrum, Pyrobacidum, Sulfolobus, Archaeoglobus, Halocarcula, Methanobacterium, Methanococcus, Methanosarcina, Methanopyrus, Pyrococcus, Picrophilus, Thermoplasma, Corynebacterium, Mycobacterium, Streptomyces, Aquifex, Porphyromonas, Chlorobium, Thermus, Bacillus, Listeria, Staphylococcus, Clostridium, Thermobacterium, Mycoplasma, Fusobacterium, Azarcus, Chromobacterium, Neisseria, Nitrosomonas, Desidfovibrio, Geobacter, Myxococcus, Campylobacter, Wolinella, Acinetobacter, Erwinia, Escherichia, Legionella, Methylococcus, Pasteurella, Photobacterium, Salmonella, Xanthomonas, Yersinia, Treponema и Thermotoga.
[698] В целом понятие "система нацеливания на нуклеиновую кислоту", используемая в настоящем изобретении, относится в совокупности к транскриптам и другим элементам, участвующим в экспрессии или направляющим активность нацеливания на нуклеиновых кислоты CRISPR-ассоциированных ("Cas") генов (также называемых в настоящем описании эффекторными белками), включая последовательности, кодирующие белок (эффекторный), нацеленный на нуклеиновую кислоту, и направляющую РНК (содержащую последовательность cr-РНК и трансактивирующую последовательность РНК (tracr-РНК) системы CRISPR/Cas) или другие последовательности и транскрипты из локуса CRISPR, нацеленного на нуклеиновые кислоты. В некоторых вариантах осуществления один или более элементов системы нацеливания на нуклеиновую кислоту получены из системы CRISPR, нацеленной на нуклеиновые кислоты, типов V/VI. В некоторых вариантах осуществления один или более элементов системы нацеливания на нуклеиновую кислоту получают из конкретного организма, включающего эндогенную систему CRISPR, нацеленную на нуклеиновые кислоты. В целом система нацеливания на нуклеиновую кислоту характеризуется элементами, которые способствуют образованию комплекса, нацеленного на нуклеиновую кислоту, на участке последовательности-мишени. В контексте формирования комплекса, нацеленного на нуклеиновую кислоту, "последовательность-мишень" относится к последовательности, для которой предназначена направляющая последовательность, и которая имеет комплементарность, необходимую для гибридизации между последовательностью-мишенью и направляющей РНК, что способствует образованию комплекса с ДНК или РНК. Полная комплементарность не обязательна, если имеется достаточная комплементарность, вызывающая гибридизацию и способствующая образованию комплекса, нацеленного на нуклеиновую кислоту. Последовательность-мишень может содержать полинуклеотиды РНК. В некоторых вариантах осуществления последовательность-мишень находится в ядре или цитоплазме клетки. В некоторых вариантах осуществления последовательность-мишень может находиться внутри органелл эукариотической клетки, например митохондрий или хлоропластов. Последовательность или матрица, которая может быть использована для рекомбинации в локусе-мишени, содержащем последовательность-мишень, упоминается как "редактирующая матрица или "редактирующая РНК" или "редактирующая последовательность". В аспектах изобретения экзогенная РНК-матрица может упоминаться как матрица редактирования. В любом аспекте изобретения рекомбинация представляет собой гомологичную рекомбинацию.
[699] Как правило, в контексте эндогенной системы нацеливания на нуклеиновую кислоту образование комплекса, нацеленного на нуклеиновую кислоту (содержащего направляющую РНК, гибридизованную с последовательностью-мишенью, в комплексе с одним или более эффекторными белками, нацеленными на нуклеиновую кислоту) приводит к расщеплению одной или обеих цепей РНК (примерно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 50 или более пар оснований) целевой последовательности. В некоторых вариантах осуществления один или более векторов, управляющих экспрессией одного или более элементов системы нацеливания на нуклеиновую кислоту, вводят в клетку, так что экспрессия элементов системы нацеливания на нуклеиновую кислоту руководит образованием комплекса, нацеленного на нуклеиновую кислоту, в одном или более участках-мишенях. Например, эффекторный белок, нацеленный на нуклеиновую кислоте, и направляющая РНК могут быть функционально связаны с отдельными регуляторными элементами на отдельных векторах. Альтернативно, два или более элемента, экспрессируемые с участием одних и тех же или разных регуляторных элементов, могут быть объединены в одном векторе с одним или более дополнительными векторами, обеспечивающими другие компоненты системы нацеливания на нуклеиновую кислоту, не включенные в первый вектор. Компоненты системы нацеливания на нуклеиновую кислоту, объединенные в одном векторе, могут быть расположены в любой подходящей ориентации, такой как один элемент, расположенный в направлении 5'-конца относительно (в восходящем направлении) или 3'-конца относительно (в нисходящем направлении) второго элемента. Кодирующая последовательность одного элемента может быть расположена на той же или противоположной цепи кодирующей последовательности второго элемента и быть ориентирована в том же или противоположном направлении. В некоторых вариантах осуществления один промотор руководит экспрессией транскрипта, кодирующего эффекторный белок, нацеленный нуклеиновую кислоту, и направляющей РНК, встроенной в одну или более интронных последовательностей (например, каждая в отдельном интроне, два или более по меньшей мере в одном интроне или все в одном интроне). В некоторых вариантах осуществления изобретения эффекторный белок, нацеленный на нуклеиновую кислоту, и направляющая РНК функционально связаны и экспрессируются с одного и того же промотора.
[700] В общем, направляющая последовательность представляет собой любую полинуклеотидную последовательность, имеющую достаточную комплементарность с полинуклеотидной последовательностью-мишенью для гибридизации с последовательностью-мишенью и прямого специфического для последовательности связывания комплекса, нацеленного на нуклеиновую кислоту, с последовательностью-мишенью. В некоторых вариантах осуществления степень комплементарности между направляющей последовательностью и соответствующей ей последовательностью-мишенью при оптимальном выравнивании с использованием подходящего алгоритма выравнивания составляет приблизительно или более чем 50%, 60%, 75%, 80%, 85%, 90%, 95%, 97,5%, 99% и более. Оптимальное выравнивание может быть определено с использованием любого подходящего алгоритма для выравнивания последовательностей, неограничивающие примеры которого, включают алгоритм Смита-Уотермана, алгоритм Нидлмана-Вунча, алгоритмы, основанные на преобразовании Берроуза-Уилера (например, Burrows Wheeler Aligner), ClustalW, Clustal X, BLAST, Novoalign (Novocraft Technologies, ELAND (iIllumina, Сан-Диего, Калифорния), SOAP (доступно на soap.genornics.org.cn) и Maq (доступно на maq.sourceforge.net). В некоторых вариантах осуществления, направляющая последовательность составляет приблизительно или больше, чем приблизительно 5, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 75 или более нуклеотидов в длину. В некоторых вариантах осуществления изобретения направляющая последовательность меньше чем приблизительно 75, 50, 45, 40, 35, 30, 25, 20, 15, 12 или менее нуклеотидов в длину. Способность направляющей последовательности управлять специфическим для последовательности связыванием нацеленного на нуклеиновую кислоту комплекса с последовательностью-мишенью может быть оценена любым подходящим способом. Например, компоненты нацеленной на нуклеиновую кислоту системы, достаточные для образования данного комплекса, включая направляющую последовательность, подлежащую тестированию, могут быть доставлены в клетку, имеющую соответствующую последовательность-мишень, например, с помощью трансфекции векторами, кодирующими компоненты нацеленной на нуклеиновые кислоты последовательности CRISPR с последующей оценкой предпочтительного расщепления в пределах или вблизи последовательности-мишени, такой как анализ Surveyor, описанный в настоящем описании. Аналогично, расщепление полинуклеотидной последовательности-мишени (или последовательности вблизи нее) может быть оценено в пробирке путем предоставления последовательности-мишени, компонентов нацеленного на нуклеиновую кислоту комплекса, включая направляющую последовательность, подлежащую тестированию, и контрольной направляющей последовательности, отличной от тестируемой направляющей последовательности, и сравнения связывания или скорости расщепления происходящего непосредственно в или вблизи последовательности-мишени между реакциями тестовой и контрольной направляющих последовательностей. Другие способы анализа также возможны и могут быть разработаны специалистами в данной области.
[701] Направляющая последовательность может быть выбрана так, чтобы нацеливать на любую последовательность-мишень. В некоторых вариантах осуществления последовательность-мишень представляет собой последовательность внутри транскрипта гена или мРНК.
[702] В некоторых вариантах осуществления последовательность-мишень представляет собой последовательность в геноме клетки.
[703] В некоторых вариантах осуществления выбирается направляющая последовательность для компактизации вторичной структуры внутри направляющей последовательности. Вторичная структура может быть определена любым подходящим алгоритмом фолдинга полинуклеотидов. Некоторые программы основаны на вычислении минимальной свободной энергии Гиббса. Примером такого алгоритма является mFold, как описано Zuker и Stiegler (Nucleic Acids Res., 9 (1981), 133-148). Другим примером алгоритма выявления фолдинга является вебсервер RNAfold, разработанный в Институте теоретической химии Венского университета, с использованием алгоритма прогнозирования центроида (см., например, AR Gruber era, 2008, Cell 106 (1): 23-24; PA Carr and GM Church, 2009, Nature Biotechnology 27 (12): 1151-62). Другие алгоритмы могут быть найдены в заявке США серийного номера TBA (номер патентного реестра 44790.11.2022, широкая ссылка В1-2013/004A); включенной в настоящее описание в качестве ссылки.
[704] В некоторых вариантах осуществления эффекторный белок, нацеленный на нуклеиновую кислоту, является частью слитого белка, содержащего один или более гетерологичных белковых доменов (например, приблизительно или более 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более доменов в дополнение к эффекторному белку, нацеленному на нуклеиновую кислоту). В некоторых вариантах осуществления эффекторный белок/фермент CRISPR является частью слитого белка, содержащего один или более гетерологичных белковых доменов (например, приблизительно или более 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, или более доменов в дополнение к ферменту CRISPR). Эффекторный белок/фермента CRISPR может содержать любую дополнительную белковую последовательность и необязательно линкерную последовательность между любыми двумя доменами. Примеры белковых доменов, которые могут быть слиты с эффекторным белком, включают, но не ограничиваются ими, метки эпитопов, последовательности репортерного гена и белковые домены, имеющие одну или более из следующих активностей: метилазная, деметилазная активность, активность активации транскрипции, активность подавления транскрипции, активность фактора терминации транскрипции, активность модификации гистонов, активность расщепления РНК и активность связывания нуклеиновых кислот. Неограничивающие примеры меток эпитопов включают метки гистидина (His), метки V5, метки FLAG, метки гемагглютинина (HA) гриппа, метки Мус, метки VSV-G и теги тиоредоксина (Trx). Примеры репортерных генов включают, но не ограничиваются следующими: глутатион-S-трансфераза (GST), пероксидаза хрена (HRP), бета-галактозидаза ацетитрансферазы хлорамфеникола (CAT), бета-глюкуронидаза, люцифераза, зеленый флуоресцентный белок (GFP), HcRed, DsRed, голубой флуоресцентный белок (CFP), желтый флуоресцентный белок (YFP) и аутофлуоресцентные белки, включая синий флуоресцентный белок (BFP). Эффекторный белок, нацеленный на нуклеиновую кислоту, может быть слит с последовательностью гена, кодирующей белок или фрагмент белка, который связывает молекулы ДНК или связывает другие клеточные молекулы, включая, но не ограничиваясь, следующими: мальтоза-связывающий белок (MBP), S-tag, слитые конструкции ДНК-связывающего домена (DBD) Lex A, слитые конструкции ДНК-связывающего домена GAL4 и слитые конструкции белка BP16 вируса простого герпеса (HSV). Дополнительные домены, которые могут быть частью слитого белка, включающего эффекторный белок, нацеленный на нуклеиновую кислоту, описаны в патентной публикации США 20110059502, включенной в настоящее описание в качестве ссылки. В некоторых вариантах осуществления меченый эффекторный белок, нацеленный на нуклеиновую кислоту, используется для идентификации местоположения последовательности-мишени.
[705] В некоторых вариантах осуществления фермент CRISPR может являться компонентом индуцируемой системы. Индуцируемый характер системы обеспечивает пространственно-временный контроль над редактированием генов или экспрессией генов с использованием энергии. Форма энергии может включать, но не ограничивается ими, электромагнитное излучение, звуковую энергию, химическую энергию и тепловую энергию. Примеры индуцируемой системы включают индуцируемые тетрациклином промоторы (Tet-On или Tet-Off), двугибридные системы активации транскрипции с малой молекулой (FKBP, ABA и т.д.), светоиндуцируемые системы (фитохром, LOV-домены или криптохром). В варианте одном осуществления изобретения фермент CRISPR может быть частью светоиндуцируемого эффектора транскрипции (LITE) для непосредственного изменения транскрипционной активности, специфичного к последовательности. Светоиндуцируемые компоненты могут включать фермент CRISPR, светочувствительный гетеродимер цитохрома (например, Arabidopsis thaliana) и домен активации/подавления транскрипции. Другие примеры индуцируемых связывающих ДНК белков и способы их использования приведены в US 61/736465 и US 61/721283 и WO 2014/018423 и US 8889418, US 8895308, US 20140186919, US 20140242700, US 20140273234, US 20140335620, WO 2014093635, которые включены в настоящее описание в качестве ссылок в полном объеме.
[706] В некоторых вариантах осуществления настоящее изобретение относится к способам, включающим доставку одного или более полинуклеотидов, таких как или один или более векторов, как описано в настоящем описании, один или более его транскриптов и/или один или более белков, транскрибируемых с них, в клетку. В некоторых аспектах изобретение, кроме того, относится к клеткам, полученным такими способами, и организмам (таким как животные, растения или грибы), содержащим или полученным из таких клеток. В некоторых вариантах осуществления эффекторный белок, нацеленный на нуклеиновую кислоту, в сочетании с (и, возможно, в составе комплекса) с направляющей РНК доставляется в клетку. Обычные вирусные и невирусные способы переноса генов могут быть использованы для введения нуклеиновых кислот в клетки млекопитающих или в целевые ткани. Такие способы могут быть использованы для введения нуклеиновых кислот, кодирующих компоненты системы нацеливания на нуклеиновую кислоту, в клетки культуры или организма. Системы доставки невирусных векторов включают ДНК-плазмиды, РНК (например, транскрипт вектора, описанного в настоящем описании), "голую" нуклеиновую кислоту и нуклеиновую кислоту в комплексе со средством доставки, таким как липосома. Системы вирусной доставки векторов включают ДНК и РНК-вирусы, которые имеют либо эписомальные, либо интегрированные геномы после доставки в клетку. Обзор способов генной терапии см. в Anderson, Science 256: 808-813 (1992); Nab el & Feigner, TIBTECH 11: 211-217 (1993), Mitani & Caskey, TIBTECH 11: 162-166 (1993); Dillon, TIBTECH 11: 167-175 (1993); Miller, Nature 357: 455-460 (1992); Van Brunt, Biotechnology 6 (10): 1149-1154 (1988); Vigne, Restorative Neurology and Neuroscience 8: 35-36 (1995), Kremer & Perricaudet, British Medical Bulletin 51 (1): 31-44 (1995); Haddada et al., В Current Topics in Microbiology and Immunology, Doerfler and Bomm (eds) (1995) и Yu et al., Gene Therapy 1: 13-26 (1994).
[707] Способы невирусной доставки нуклеиновых кислот включают липофекцию, нуклеофекцию, микроинъекцию, генные пушки, виросомы, липосомы, иммунолипосомы, поликатионы или конъюгаты липидов с нуклеиновыми кислотами, "голую" ДНК, искусственные вирионы и усиленное агентом поглощение ДНК. Липофекция описана, например, в патентах США №5049386, 4946787; и 4897355), и липофекционные реагенты продаются на коммерческой основе (например, Transfectam™ и Lipofectin™). Катионные и нейтральные липиды, которые пригодны для эффективного распознавания рецепторов липофекции полинуклеотидов, включают таковые Feigner, WO 91/17424; WO 91/16024. Доставка может осуществляться в клетки (например, in vitro или ex vivo) или ткани-мишени (например, in vivo).
[708] Получение комплексов липидов с нуклеиновыми кислотами, включая целевые липосомы, такие как иммунолипидные комплексы, хорошо известно специалисту в данной области (см., например, Crystal, Science 270: 404-410 (1995), Blaese et al., Cancer Gene Ther. 2: 291-297 (1995), Behr et at., Bioconjugate Chem., 5: 382-389 (1994), Remy et al., Bioconjugate Chem., 5: 647-654 (1994), Gao et al., Gene Therapy 2: 710-722 (1995), Ahmad et al., Cancer Res. 52: 4817-4820 (1992), патенты США №№4186183, 4217344, 4235871, 4261975, 4485054, 4501728, 4774085, 4837028 и 4946787).
[709] В применении РНК или ДНК-вирусных систем для доставки нуклеиновых кислот используются современные способы нацеливания вируса на конкретные клетки в организме и передачи вирусного генома в ядро. Вирусные векторы могут вводиться непосредственно пациентам (in vivo) или их можно использовать для обработки клеток in vitro, а модифицированные клетки могут быть введены пациентам (ex vivo). Обычные вирусные системы могут включать ретровирусные, лентивирусные, аденовирусные, аденоассоциированные и вирусные вирусы простого герпеса для переноса генов. Встраивание в геном хозяина возможно с помощью способов переноса генов ретровирусов, лентивирусов и аденоассоциированных вирусов, часто приводящих к длительной экспрессии введенного трансгена. Кроме того, высокая эффективность трансдукции наблюдалась во многих других типах клеток- и тканей-мишеней.
[710] Тропизм ретровируса может быть изменен путем включения чужеродных белков оболочки, что позволяет расширить потенциальную популяцию клеток, которые могут стать мишенью генного воздействия. Лентивирусные векторы представляют собой ретровирусные векторы, которые способны трансформировать или инфицировать не делящиеся клетки и, как правило, имеют высокие вирусные титры. Поэтому выбор ретровирусной системы переноса генов будет зависеть от ткани-мишени. Ретровирусные векторы состоят из цис-действующих длинных концевых повторов с упаковочной емкостью до 6-10 т.п.н. чужеродной последовательности. Минимальные цис-действующие LTR достаточны для репликации и упаковки векторов, которые затем используются для интеграции терапевтического гена в клетку-мишень для обеспечения постоянной трансгенной экспрессии. Широко используемые ретровирусные векторы включают векторы, основанные на вирусе лейкемии мышей (MuLV), вирусе лейкемии гиббонов (GaLV), вирусе иммунодефицита обезьян (SIV), вирусе иммунодефицита человека (ВИЧ) и их комбинациях (см., например, Buchscher et al., J. Virol, 66: 2731-2739 (1992), Johann et al., J. Virol. 66: 1635-1640 (1992), Sommnerfelt et al., Virol. 176: 58-59 (1990), Wilson et al., J. Virol, 63: 2374-2378 (1989), Miller et al., J. Virol. 65: 2220-2224 (1991), PCT/US94/05700). В тех вариантах, где предпочтительна временная экспрессия, могут быть использованы аденовирусные системы. Аденовирусные векторы способны к очень высокой эффективности трансдукции во многих типах клеток и не требуют деления клеток. С такими векторами был получен высокий титр и уровни экспрессии. Этот вектор может быть получен в больших количествах в относительно простой системе. Векторы на основе аденоассоциированных вирусов ("AAV") также могут быть использованы для трансдукции клеток целевыми нуклеиновыми кислотами, например, при продуцировании in vitro нуклеиновых кислот и пептидов, а также для процедур генной терапии in vivo и ex vivo (см., например, West et al., Virology 160: 38-47 (1987), US 4797368, WO 93/24641, Kotin, Human Gene Therapy 5: 793-801 (1994), Muzyczka, J. Clin. Invest.: 1351 (1994). Конструирование рекомбинантных векторов AAV описано в ряде публикаций, включая патент США №5173414, Tratschin et al. Mol. Cell, Biol., 5: 3251-3260 (1985), Tratschin, et al., Mol. Cell, Biol., 4: 2072-2081 (1984), Hermonat & Muzyczka, PNAS 81: 6466-66470 (1984) и Samulski et al., J. Virol. 63: 03822-3828 (1989).
Модели генетических и эпигенетических условий
[711] Способ по изобретению может быть использован для создания растения, животного или клетки, которые могут быть использованы для моделирования и/или изучения генетических или эпигенетических условий, представляющих интерес, например, посредством модели представляющих интерес мутаций или заболеваний. Как используют в настоящем описании, термин "заболевание" относится к заболеванию, расстройству или симптому у индивида. Например, способ по изобретению может быть использован для создания животного или клетки, которые включают модификацию в одной или более последовательностях нуклеиновых кислот, связанных с заболеванием, или растение, животное или клетку, в которых экспрессия одной или более последовательностей нуклеиновых кислот, связанных с заболеванием, изменена. Такая последовательность нуклеиновых кислот может кодировать связанную с заболеванием последовательность белка или может быть связанной с заболеванием последовательностью контроля. Соответственно, понятно, что в вариантах осуществления изобретения растение, индивид, пациент, организм или клетка могут быть не являющимися человеком пациентом, организмом или не происходящей от человека клеткой. Таким образом, изобретение относится к растению, животному или клетке, полученным способами по настоящему изобретению, или к их потомству. Потомство может быть клоном произведенного растения или животного или может быть результатом полового размножения путем скрещивания с другими особями одного и того же вида для внедрения других желательных признаков в их потомство. Клетка может находиться in vivo или ex vivo в случае многоклеточных организмов, особенно животных или растений. В случае, когда клетка культивируется, клеточная линия может быть создана, если соответствующие условия культивирования удовлетворяются, и предпочтительно, если клетка подходяще приспособлена для этой цели (например, стволовая клетка). Также предусматриваются бактериальные клеточные линии, полученные с соответствии с изобретением. Таким образом, клеточные линии также предусматриваются.
[712] В некоторых способах модель болезни может быть использована для изучения эффектов мутаций на животное или клетку, развития и/или прогрессирования заболевания с использованием мер, обычно используемых при изучении этого заболевания. Альтернативно, такая модель болезни полезна для изучения влияния фармацевтически активных соединений на заболевание.
[713] В некоторых способах модель болезни может быть использована для оценки эффективности потенциальной стратегии генной терапии. То есть связанный с болезнью ген или полинуклеотид может быть модифицирован таким образом, что развитие заболевания и/или его прогрессирование ингибируются или уменьшаются. В частности, способ включает модификацию связанного с заболеванием гена или полинуклеотида таким образом, что образуется измененный белок и, как результат, животное или клетка имеют измененный ответ. Соответственно, в некоторых способах генетически модифицированное животное можно сравнить с животным, предрасположенным к развитию заболевания, что даст возможность оценить влияние генной терапии.
[714] В другом варианте осуществления настоящее изобретение относится к способам разработки биологически активного агента, который изменяет сигнал клеток, связанных с геном болезни. Способ включает приведение в контакт испытуемого соединения с клеткой, содержащей один или более векторов, управляющих экспрессией одного или более ферментов CRISPR, и последовательность прямого повтора, связанную с направляющей последовательностью; и обнаружение изменения в показаниях, которое указывает на уменьшение или увеличение сигнала клетки, связанного, например, с мутацией в гене болезни, содержащемся в клетке.
[715] Модель клетки или модель на животных может быть создана в сочетании со способом изобретению для диагностики изменения клеточной функции. Такая модель может быть использована для изучения влияния последовательности генома, модифицированной комплексом CRISPR по изобретению, на интересующую клеточную функцию. Например, модель клеточной функции может использоваться для изучения влияния модифицированной последовательности генома на внутриклеточную передачу сигнала или внеклеточную передачу сигнала. Альтернативно, модель клеточной функции может использоваться для изучения эффектов модифицированной последовательности генома на сенсорное восприятие. В некоторых таких моделях модифицирована одна или более геномных последовательностей, связанных с сигнальным биохимическим путем в модели.
[716] Было исследовано несколько моделей заболеваний. К ним относятся исследование de novo генов риска аутизма CHD8, KATNAL2 и SCN2A; и гена синдромного аутизма (синдром Ангельмана) UBE3A. Конечно, эти гены и результирующие модели аутизма предпочтительнее, но служат для того, чтобы показать широкую применимость изобретения для генов и соответствующих моделей.
[717] Измененная экспрессия одной или более геномных последовательностей, связанных с сигнальным биохимическим путем, может быть определена путем анализа разности уровней мРНК соответствующих генов между тестовой модельной клеткой и контрольной клеткой, когда они контактируют с кандидатом. Альтернативно, дифференциальная экспрессия последовательностей, связанных с сигнальным биохимическим путем, определяется путем обнаружения различий в количестве кодируемого полипептида или генного продукта.
[718] Для анализа индуцированного агентом изменения уровня транскриптов мРНК или соответствующих полинуклеотидов нуклеиновую кислоту, содержащуюся в образце, сначала экстрагируют в соответствии со стандартными способами в данной области. Например, мРНК может быть выделена с использованием различных литических ферментов или химических растворов в соответствии с методиками, изложенными в Sambrook et al. (1989) или экстрагирована путем связывания нуклеиновой кислоты со смолами в соответствии с прилагаемыми инструкциями производителей. Затем мРНК, содержащуюся в образце экстрагированной нуклеиновой кислоты, выявляют с помощью способов амплификации или обычных анализов гибридизации (например, нозерн-блоттинга) в соответствии со способами, широко известными в данной области или на основе способов, описанных в настоящем описании.
[719] Для целей настоящего изобретения амплификация означает любой способ, использующий праймер и полимеразу, способную реплицировать последовательность-мишень с достаточной точностью. Амплификацию можно проводить с помощью природных или рекомбинантных ДНК-полимераз, таких как ДНК-полимераза TaqGold™, T7, фрагмент Кленова ДНК-полимеразы E.coli и обратная транскриптаза. Предпочтительным способом амплификации является ПЦР. В частности, выделенная РНК может быть подвергнута анализу с использованием обратной транскрипции, которая сопряжена с количественной полимеразной цепной реакцией (ОТ-РЦР), чтобы количественно определить уровень экспрессии последовательности, связанной с сигнальным биохимическим путем.
[720] Определение уровня экспрессии гена может проводиться в реальном времени в анализе с использованием амплификации. В одном из аспектов амплифицированные продукты могут быть непосредственно визуализированы с помощью флуоресцентных связывающих ДНК агентов, включая, но не ограничиваясь ими: интеркаляторы ДНК и связывающиеся с желобками ДНК агенты. Поскольку количество интеркаляторов, включенных в молекулы двухцепочечной ДНК, как правило, пропорционально количеству амплифицированных продуктов ДНК, удобно определять количество амплифицированных продуктов путем количественной оценки флуоресценции интеркалировавшего красителя с использованием стандартно применяемых оптических приборов, используемых в данной области. ДНК-связывающие красители, подходящие для применения с этой целью, включает SYBR green, SYBR blue, DAPI, пропидия йодид, hoechst, SYBR gold, бромид этидия, акридиновые красители, дауномицин, хлорохин, дистамицин D, хромомицин, митрамицин, полипиридины рутения, антрамицин и т.п.
[721] В другом аспекте в реакции амплификации могут быть использованы другие флуоресцентные метки, такие как специфические для последовательности зонды для облегчения обнаружения и количественного определения амплифицированных продуктов. Количественная амплификация на основе использования зонда зависит от специфического для последовательности определения желаемого продукта амплификации. Она использует флуоресцентные целеспецифичные зонды (например, зонды TaqMan®), что приводит к повышению специфичности и чувствительности. Способы осуществления количественной амплификации на основе использования зонда хорошо известны в данной области и описаны в патенте США №5210015.
[722] В еще одном варианте аспекте могут быть выполнены обычные анализы гибридизации с использованием зондов гибридизации, гомологичных последовательностям, связанным с сигнальным биохимическим путем. Обычно допускается формирование зондами стабильных комплексов с последовательностями, связанными с сигнальным биохимическим путем, содержащимися в биологическом образце, полученном от исследуемого индивидуума, в ходе реакции гибридизации. Квалифицированному специалисту в данной области должно быть понятно, что, когда антисмысловая последовательность используется в нуклеиновой кислоте-зонде, полинуклеотиды-мишени, представленные в образце, выбирают так, чтобы они были комплементарными антисмысловым последовательностям нуклеиновых кислот. Напротив, когда нуклеотидный зонд является смысловой последовательностью нуклеиновой кислоты, в качестве полинуклеотида-мишени выбирают комплементарную к последовательностям смысловую последовательность нуклеиновой кислоты.
[723] Гибридизация может выполняться в условиях, различающихся по уровню жесткости. Подходящие условия гибридизации для практики настоящего изобретения таковы, что взаимодействие при распознавании зондом последовательностей, ассоциированных с сигнальным биохимическим путем, является достаточно специфичным и достаточно стабильным. Условия, которые повышают жесткость условий реакции гибридизации, широко известны и опубликованы в данной области. См., например, (Sambrook, et al., (1989), Nonradioactive In Situ Hybridization Application Manual, Boehringer Mannheim, second edition). Анализ гибридизации может быть проведен с использованием зондов, иммобилизованных на любом твердом носителе, включая, но не ограничиваясь, нитроцеллюлозу, стекло, силикон и множество генных матриц. Является предпочтительным проведение анализа гибридизации на генных чипах высокой плотности, как описано в патенте США №5445934.
[724] Для удобного обнаружения комплексов зонд-мишень, образующихся во время анализа гибридизации, нуклеотидные зонды могут быть конъюгированы с поддающейся обнаружению меткой. Поддающиеся обнаружению метки, подходящие для использования в рамках настоящего изобретения, включают любую конструкцию, детектируемую фотохимическими, биохимическими, спектроскопическими, иммунохимическими, электрическими, оптическими или химическими способами. В данной области известно большое количество подходящих поддающихся обнаружению меток, которые включают флуоресцентные или хемилюминесцентные метки, метки радиоактивного изотопа, ферментативные или другие лиганды. В предпочтительных вариантах осуществления, вероятно, может быть желательным использовать флуоресцентную метку или метку фермента, такую как дигоксигенин, β-галактозидаза, уреаза, щелочная фосфатаза или пероксидаза, комплекс авидин/биотин.
[725] Способы обнаружения, используемые для обнаружения или количественной оценки гибридизации, обычно зависят от выбора метки. Например, радиоактивные метки могут быть обнаружены с использованием фотопленки или прибора Phosphoimager. Флуоресцентные маркеры могут быть обнаружены и количественно определены с помощью фотоприемника для обнаружения испускаемого света. Ферментативные метки обычно обнаруживают путем обеспечения фермента субстратом и измерения количества полученного продукта реакции, создаваемого воздействием фермента на субстрат; и, наконец, колориметрические метки обнаруживаются путем простой визуализации цветной метки.
[726] Индуцированное агентом изменение экспрессии последовательностей, связанных с сигнальным биохимическим путем, также может быть определено путем изучения соответствующих продуктов гена. Определение уровня белка обычно включает: а) проведение реакции белка, содержащегося в биологическом образце, с агентом, который специфически связывается с белком, ассоциированным с сигнальным биохимическим путем; и (b) идентификацию любого образовавшегося комплекса агент-белок. В одном аспекте данного варианта осуществления агент, который специфически связывает белок, связанный с сигнальным биохимическим путем, представляет собой антитело, предпочтительно моноклональное антитело.
[727] Реакцию проводят путем связывания агента с образцом белков, ассоциированных с сигнальным биохимическим путем, полученным из пробных образцов, в условиях, которые позволяют образование комплекса агентом и белками, ассоциированными с сигнальным биохимическим путем. Образование комплекса может быть обнаружено прямо или косвенно в соответствии со стандартными методиками в данной области. При использовании способа прямого обнаружения агенты снабжаются поддающейся обнаружению меткой, и непрореагировавшие агенты могут быть удалены из комплекса; количество неиспользованной метки тем самым позволяет определить количество образовавшегося комплекса. Для такого способа предпочтительно выбирать метки, которые остаются связанными с агентом даже при жестких условиях промывания. Предпочтительно, чтобы метка не мешала реакции связывания. В альтернативном варианте процедура косвенного обнаружения может использовать агент, который содержит метку, введенную либо химическим, либо ферментативным путем. Желательная метка обычно не препятствует связыванию или стабильности полученного комплекса полипептида и агента. Однако метка обычно предназначена для того, чтобы быть доступной для эффективного связывания антителом и, следовательно, генерировать распознаваемый сигнал.
[728] В данной области известно большое количество меток, подходящих для определения уровней белка. Неограничивающие примеры включают радиоизотопы, ферменты, коллоидные металлы, флуоресцентные соединения, биолюминесцентные соединения и хемилюминесцентные соединения
[729] Количество комплексов агент-полипептид, образующихся во время реакции связывания, может быть количественно определено стандартными количественными способами анализа. Как проиллюстрировано выше, образование комплекса агент-полипептид может быть непосредственно измерено по количеству метки, оставшейся в участке связывания. В альтернативном варианте белок, связанный с сигнальным биохимическим путем, тестируют на его способность конкурировать с меченым аналогом за сайты связывания специфического агента. В этом конкурентном анализе количество захваченной метки обратно пропорционально количеству белковых последовательностей, ассоциированных с сигнальным биохимическим путем, присутствующих в тестируемом образце.
[730] Ряд способов анализа белка, основанных на общих принципах, изложенных выше, доступен в данной области. Они включают, но не ограничиваются ими, радиоиммунные способы анализа, ELISA (иммунорадиометрические анализ с ферментным связыванием), "сэндвич-иммуноанализ", иммунорадиометрический анализ, иммунологический анализ in situ (с использованием, например, коллоидного золота, фермента или радиоизотопной метки), анализ с использованием вестерн-блоттинга, анализ иммунопреципитации, иммунофлуоресцентный анализ и SDS-PAGE.
[731] Антитела, которые специфически распознают или связывают с белки, связанные с сигнальным биохимическим путем, являются предпочтительными для проведения вышеупомянутых анализов белка. При желании можно использовать антитела, которые распознают конкретный тип посттрансляционных модификаций (например, индуцирбельные модификации сигнального биохимического пути). Посттрансляционные модификации включают, но не ограничиваются ими: гликозилирование, липидирование, ацетилирование и фосфорилирование. Эти антитела могут быть приобретены у коммерческих поставщиков. Например, доступны антитела к антифосфотирозину, которые специфически распознают тирозин-фосфорилированные белки, ряда поставщиков, включая Invitrogen и Perkin Elmer. Антитела к антифосфотирозину особенно полезны для обнаружения белков, которые по-разному фосфорилируются по остаткам тирозина в ответ на стресс ER. Такие белки включают, но не ограничиваются этим, эукариотический фактор инициации трансляции 2 альфа (eIF-2a). Альтернативно, эти антитела могут быть получены с использованием традиционных технологий поликлональных или моноклональных антител путем иммунизации животного-хозяина или клетки, продуцирующей антитела, целевым белком, который демонстрирует желаемую посттрансляционную модификацию.
[732] При практическом осуществлении способа введения в индивида может быть желательным выявить паттерн экспрессии белка, связанного с сигнальным биохимическим путем в различных тканях организма, в разных типах клеток и/или в разных субклеточных структурах. Эти исследования могут быть выполнены с использованием специфичных для ткани, специфичных для клеток или субклеточных структур антител, способных связываться с маркерами белка, которые предпочтительно экспрессируются в определенных тканях, типах клеток или субклеточных структурах.
[733] Измененная экспрессия гена, связанного с сигнальным биохимическим путем, также может быть определена путем изучения изменения активности генных продуктов в сравнении с контрольной клеткой. Анализ вызванного агентом изменения активности белка, связанного с сигнальным биохимическим путем, будет зависеть от биологической активности и/или пути передачи сигнала, который исследуется. Например, когда белок является киназой, изменение его способности фосфорилировать субстрат (субстраты) ниже в том же пути можно определить с помощью различных анализов, известных в данной области. Типичные способы анализа включают, однако не ограничиваются такими как: иммуноблоттинг и иммунопреципитацию антителами, такими как антитела к антифосфотирозину, которые распознают фосфорилированные белки. Кроме того, активность киназы может быть обнаружена с помощью высокопроизводительных хемилюминесцентных способов анализа, таких как AlphaScreen™ (доступный от Perkin Elmer) и eTag™ (Chan-Hui, et al., (2003) Clinical Immunology 111: 162-174).
[734] Если белок, связанный с сигнальным биохимическим путем, является частью сигнального каскада, приводящего к колебаниям внутриклеточного рН, в качестве репортерных молекул можно использовать чувствительные к рН молекулы, такие как флуоресцентные рН-индикаторы. В другом примере, где белок, связанный с сигнальным биохимическим путем, является ионным каналом, можно контролировать флуктуации мембранного потенциала и/или концентрации внутриклеточных ионов. Ряд коммерческих наборов и высокопроизводительных устройств особенно подходит для быстрого и надежного скрининга модуляторов ионных каналов. Типичные устройства включают FLIPR™ (Molecular Devices, Inc.) и VIPR (Aurora Biosciences). Эти приборы способны одновременно обнаруживать реакции в более чем 1000 лунках образцов микропланшета и обеспечивать измерение и функциональные данные в режиме реального времени в течение секунд или даже миллисекунд.
[735] При использовании любого из описанных в настоящем описании способов подходящий вектор может быть введен в клетку или эмбрион с использованием одного или более способов, известных в данной области, включая, но не ограничиваясь этим, микроинъекции, электропорацию, образование пор с использованием ультразвука, биолистику, опосредованную фосфатом кальция трансфекцию, катионную трансфекцию, липосомную трансфекцию, трансфекцию с использованием дендримеров, трансфекцию с использованием теплового шока, трансфекцию-нуклеофекцию, магнитофекцию, липофекцию, импалефекцию, оптическую трансфекцию, захват нуклеиновых кислот патентованным агентом и доставку с использованием липосом, иммунолипосом, вирусом и искусственных вирионов. В некоторых способах вектор вводится в эмбрион путем микроинъекции. Вектор или векторы могут быть введены посредством микроинъекции в ядро или цитоплазму эмбриона. В некоторых способах вектор или векторы могут быть введены в клетку путем нуклеофекции.
[736] Полинуклеотид-мишень комплекса CRISPR может быть любым полинуклеотидом, эндогенным или экзогенным для эукариотической клетки. Например, полинуклеотид-мишень может представлять собой полинуклеотид, который находится в ядре эукариотической клетки. Полинуклеотид-мишень может представлять собой последовательность, кодирующую генный продукт (например, белок) или некодирующую последовательность (например, регуляторный полинуклеотид или мусорную ДНК).
[737] Примеры полинуклеотидов-мишеней включают последовательность, связанную с сигнальным биохимическим путем, например, сигнальным биохимическим путем, ассоциированным с геном или полинуклеотидом. Примеры полинуклеотидов-мишеней включают связанный с заболеванием ген или полинуклеотид. Понятие ген или полинуклеотид, "ассоциированный с заболеванием" относится к любому гену или полинуклеотиду, который производит продукты транскрипции или трансляции на аномальном уровне или в аномальной форме в клетках, полученных из тканей, пораженных болезнью, по сравнению с контрольными тканями или клетками, не пораженными заболеванием. Это может быть ген, который экспрессируется на аномально высоком уровне; это может быть ген, который экспрессируется на аномально низком уровне, причем изменения уровня экспрессии коррелируют с возникновением и/или прогрессированием заболевания. Ассоциированный с заболеванием ген также относится к гену, имеющему мутацию(и) или генетическую вариацию, которая непосредственно ответственна или находится в неравновесной взаимосвязи с геном (генами), который ответственен за этиологию заболевания. Продукты транскрипции или трансляции могут быть известны или неизвестны и могут быть на нормальном или аномальном уровне.
[738] Полинуклеотид-мишень комплекса CRISPR может быть любым полинуклеотидом, эндогенным или экзогенным, в эукариотической клетке. Например, полинуклеотид-мишень может быть полинуклеотидом, находящимся в ядре эукариотической клетки. Полинуклеотид-мишень может представлять собой последовательность, кодирующую генный продукт (например, белок) или некодирующую последовательность (например, регуляторный полинуклеотид или мусорную ДНК). Не ограничиваясь теорией, считается, что последовательность-мишень должна быть связана с PAM (мотив, находящийся вблизи протоспесера); то есть короткой последовательностью, распознаваемой комплексом CRISPR. Точные требования к последовательности и длине для последовательности PAM различаются в зависимости от используемого фермента CRISPR, но последовательность PAM обычно находится в 2-5 парах оснований от протоспейсера (то есть последовательности-мишени). Примеры последовательностей PAM приведены в разделе примеров ниже, и квалифицированный специалист в данной области сможет идентифицировать дальнейшие последовательности PAM для использования с данным ферментом CRISPR.
[739] Полинуклеотид-мишень комплекса CRISPR может включать ряд ассоциированных с заболеванием генов и полинуклеотидов, а также генов и полинуклеотидов, ассоциированных с сигнальными биохимическими путями, как указано в предварительных патентных заявках США 61/736527 и 61/748427, обе под названием "SYSTEMS METHODS AND COMPOSITIONS FOR SEQUENCE MANIPULATION", поданных 12 декабря 2012 года и 2 января 2013 года, соответственно, и заявке РСТ PCT/US2013/074667, озаглавленной "DELIVERY, ENGINEERING AND OPTIMIZATION OF SYSTEMS, METHODS AND COMPOSITIONS FOR SEQUENCE MANIPULATION AND THERAPEUTIC APPLICATIONS", поданной 12 декабря 2013 года, содержание каждой из которых включено в настоящее описание в качестве ссылки в полном объеме.
[740] Примеры полинуклеотидов-мишеней включают последовательность, ассоциированную с сигнальным биохимическим путем, например, ген или нуклеотид, ассоциированный с сигнальным биохимическим путем. Примеры полинуклеотидов-мишеней включают ассоциированный с заболеванием ген или полинуклеотид. Ген или полинуклеотид, "ассоциированный с заболеванием", относится к любому гену или полинуклеотиду, который дает продукты транскрипции или трансляции на аномальном уровне или в аномальной форме в клетках, полученных из тканей, пораженных заболеванием, по сравнению с контрольными тканями или клетками, не пораженными заболеванием. Это может быть ген, который экспрессируется на аномально высоком уровне; это может быть ген, который экспрессируется на аномально низком уровне, когда измененная экспрессия коррелирует с возникновением и/или прогрессированием заболевания. "Ассоциированный с заболеванием ген" также относится к гену, обладающему мутацией (мутациями) или генетической вариацией, которая непосредственно ответственна или находится в неравновесии по сцеплению с геном(ами), который отвечает за этиологию заболевания. Транскрибированные или транслированные продукты могут быть известны или неизвестны и могут быть на нормальном или ненормальном уровне.
Полногеномный скрининг посредством нокаута
[741] Экспериментальные белковые комплексы CRISPR, описанные в настоящем описании, могут быть использованы для проведения эффективного и экономически эффективного функционального геномного скрининга. При таком скрининге можно использовать геномные библиотеки, основанные на эффекторном белке CRISPR. Такой скрининг и библиотеки могут обеспечить определение функций генов, клеточных путей, в которые вовлечены гены, и то, как какое-либо изменение экспрессии гена может привести к определенному биологическому процессу. Преимущество настоящего изобретения состоит в том, что система CRISPR позволяет избежать нецелевого связывания и возникающих в результате неспецифических эффектов. Это достигается с использованием систем, организованных с высокой степенью специфичности последовательности к ДНК-мишени. В предпочтительных вариантах осуществления изобретения комплексы эффекторных белков CRISPR представляют собой комплексы эффекторных белков C2c2.
[742] В вариантах осуществления изобретения полногеномная библиотека может содержать множество направляющих РНК для С2с2, как описано в настоящем описании, включая направляющие последовательности, которые способны нацеливать на множество последовательностей-мишеней во множестве геномных локусов в популяции эукариотических клеток. Популяция клеток может представлять собой популяцию эмбриональных стволовых (ES) клеток. Последовательность-мишень в геномном локусе может быть некодирующей последовательностью. Некодирующая последовательность может быть интронной, регуляторной последовательностью, сайтом сплайсинга, 3-'UTR, 5'-UTR или сигналом полиаденилирования. Функция одного или более генных продуктов может быть изменена путем нацеливания. Нацеливание может привести к нокауту функции гена. Нацеливание на генный продукт может включать более одной направляющей РНК. Нацеливание на генный продукт может быть осуществлено посредством 2, 3, 4, 5, 6, 7, 8, 9 или 10 направляющих РНК, предпочтительно от 3 до 4 на ген. Нецелевые модификации могут быть сведены к минимуму за счет использования ступенчатых разрывов двойной цепи, образуемых эффекторными белковыми комплексами C2c2, или с использованием способов, аналогичных тем, которые используются в системах CRISPR-Cas9 (см., например, DNA targeting specificity of RNA-guided Cas9 nucleases. Hsu, P., Scott, D., Weinstein, J., Ran, FA., Konermann, S., Agarwala, V., Li, Y., Fine, E., Wu, X., Shalem, O., Cradick, TX, Marraffmi, LA., Bao, G., & Zhang, F. Nat Biotechnol doi: 10.1038/nbt.2647 (2013)), включенной в настоящее описание в качестве ссылки. Нацеливание может охватывать приблизительно 100 или более последовательностей. Нацеливание может охватывать приблизительно 1000 или более последовательностей. Нацеливание может составлять приблизительно 20000 или более последовательностей. Нацеливание может охватывать весь геном. Нацеливание может охватывать ряд последовательностей-мишеней, ориентированных на соответствующий или желательный процесс. Процесс может быть иммунным процессом. Процесс может быть процессом клеточного деления.
[743] Один из вариантов изобретения охватывает геномную или транскриптомную библиотеку, которая может содержать множество направляющих РНК для C2c2, которые могут содержать направляющие последовательности, которые могут нацеливать на множество последовательностей-мишеней во множестве (геномных) локусов, приводя к нокауту/нокдауну функции гена. Эта библиотека может потенциально содержать направляющие РНК, которые нацелены на каждый ген в геноме организма.
[744] В некоторых вариантах осуществления изобретения организм или индивид является эукариотическим организмом (включая млекопитающее, включая человека), или эукариотическим организмом, не являющимся человеком, или животным, не являющимся человеком, или млекопитающим, не являющимся человеком. В некоторых вариантах осуществления организм или индивид является животным, не являющимся человеком, и может быть членистоногим, например насекомым, или может быть нематодой. В некоторых способах изобретения организм или индивид является растением. В некоторых способах по изобретению организм или субъект представляет собой млекопитающее или млекопитающее, не являющееся человеком. Млекопитающее, не являющееся человеком, может быть, например, грызуном (предпочтительно мышью или крысой), копытным или приматом. В некоторых способах по изобретению организм или субъект является водорослью, включая микроводоросли, или грибом.
[745] Нокаут/нокдаун функции гена может включать введение в каждую клетку в популяции клеток векторной системы из одного или более векторов, содержащих сконструированную, не встречающуюся в природе систему, содержащую I. эффекторный белок C2c2, и II. одну или более направляющих РНК, где компоненты I и II могут находиться на одном или разных векторах системы, встраивающих компоненты I и II в каждую клетку, причем направляющая последовательность нацелена на уникальный ген в каждой клетке, где эффекторный белок C2c2 является функционально связанным с регуляторным элементом, где при транскрипции направляющая РНК, содержащая направляющую последовательность, обеспечивает последовательность-специфическое связывание системы эффекторного белка C2c2 с последовательностью-мишенью, соответствующей геномным локусам уникального гена, вызывая расщепление РНК, соответствующей указанному геномному локусу, эффекторным белком C2c2, и подтверждение различных случаев нокдауна во множестве уникальных генов в каждой клетке популяции, тем самым, получая библиотеку клеток с нокдауном гена. Изобретение подразумевает, что популяция клеток представляет собой популяцию эукариотических клеток, и в предпочтительном варианте осуществления популяция клеток представляет собой популяцию эмбриональных стволовых клеток (ES).
[746] Один или более векторов могут быть плазмидными векторами. Вектор может представлять собой один вектор, содержащий эффекторный белок C2c2, одноцепочечную направляющую РНК и необязательно селективный маркер для клеток-мишеней. Без связи с теорией, одновременная доставка эффекторного белка C2c2 и одноцепочечной направляющей РНК с помощью одного вектора позволяет применять этот способ к любому типу клеток-мишеней без необходимости сначала получать клеточные линии, которые экспрессируют эффекторный белок C2c2. Регулирующим элементом может быть индуцибельный промотор. Индуцируемый промотор может быть промотором, индуцируемым доксициклином. В некоторых способах по изобретению экспрессия направляющей последовательности находится под контролем промотора Т7 и направляется экспрессией Т7-полимеразы. Подтверждение различных случаев нокдауна может быть осуществлено полнотранскриптомным секвенированием. Мутация нокаута может быть достигнута в 100 или более уникальных генах. Нокдаун может быть достигнут в 1000 или более уникальных генах. Нокдаун может быть достигнут в 20000 или более уникальных генов. Нокдаун может быть достигнут и во всем геноме. Нокдаун функции гена может быть достигнут во множестве уникальных генов, которые функционируют в определенном физиологическом процессе или состоянии. Процесс или состояние может быть иммунным процессом или состоянием. Процесс или состояние может быть процессом или состоянием деления клеток.
[747] Изобретение также относится к наборам, включающим обширные библиотеки транскриптомов, упомянутые в настоящем описании. Набор может содержать один контейнер, содержащий векторы или плазмиды, содержащие библиотеку по изобретению. Набор может также содержать панель, содержащую отобранный комплект уникальных направляющих РНК системы эффекторных белков C2c2, содержащих направляющие последовательности из библиотеки по изобретению, причем отобранный комплект соответствует конкретному физиологическому состоянию. В рамках изобретения подразумевается, что нацеливанию подвергается приблизительно 100 или более последовательностей, приблизительно 1000 или более последовательностей или приблизительно 20000 или более последовательностей или весь транскриптом. Кроме того, панель последовательностей-мишеней может быть сфокусирована на соответствующем или желательном процессе, таком как иммунный процесс или деление клеток.
[748] В дополнительном аспекте изобретения эффекторный белок C2c2 может содержать одну или более мутаций и может быть использован в качестве общего РНК-связывающего белка с или без слияния с функциональным доменом. Мутации могут быть искусственно введенными мутациями или мутациями приобретения или потери функции. Мутации были охарактеризованы, как описано в настоящем описании. В одном аспекте изобретения функциональный домен может быть доменом активации транскрипции, который может быть VP64. В других аспектах изобретения функциональный домен может быть доменом подавления транскрипции, который может быть KRAB или SID4X. Другие аспекты изобретения относятся к мутантному эффекторному белку C2c2, слитому с доменами, которые включают, но не ограничиваются, ими: активатор транскрипции, репрессор, рекомбиназу, транспозазу, ремоделирующий гистон, деметилазу, ДНК-метилтрансферазу, криптохром, светоиндуцируемый/контролируемый домен или химически индуцируемый/контролируемый домен. Некоторые способы по изобретению могут включать индукцию экспрессии генов-мишеней. В одном варианте осуществления индукция экспрессии путем нацеливания на множество последовательностей-мишеней во множестве геномных локусов в популяции эукариотических клеток осуществляется с использованием функционального домена.
[749] В практике настоящего изобретения, использующего комплексы эффекторного белка C2c2, могут использоваться способы, в которых используются системы CRISPR-Cas9, и может быть приведена ссылка на:
[750] Genome-Scale CRISPR-Cas9 Knockout Screening in Human Cells. Shalem, O., Sanjana, NE., Hartenian, E., Shi, X., Scott, DA., Mikkelson, T., Heckl, D., Ebert, BE., Root, DE., Doench, JG., Zhang, F. Science Dec 12. (2013). Опубликовано в окончательной редакции как: Science. 2014 Jan 3; 343(6166): 84-87
[751] В Shalem et al. описан новый способ исследования функции гена в масштабе всего генома. Их исследования показали, что доставка полногеномной библиотеки с нокаутом CRISPR-Cas9 (GeCKO), нацеленной на 18080 генов с 64751 уникальными направляющими последовательностями, позволила скрининг как с отрицательной, так и с положительной селекцией в клетках человека. Во-первых, авторы показали использование библиотеки GeCKO для идентификации генов, необходимых для жизнеспособности клеток в злокачественных и плюрипотентных стволовых клетках. Затем, с использованием модели меланомы, авторы провели скрининг генов, утрата которых связана с резистентностью к вемурафенибу, препарату, который ингибирует мутантную протеинкиназу BRAF. Их исследования показали, что кандидаты с самым высоким рейтингом включают ранее подтвержденные гены NF1 и MED 12, также как и новые высокие результаты: NF2, CUL3, TADA2B и TADA1. Авторы наблюдали высокий уровень согласованности между независимыми направляющими РНК, нацеленными на один и тот же ген, и высокий показатель подтверждения высоких результатов, и, таким образом, продемонстрировали перспективность скрининга генома с помощью Cas9.
[752] См. также патент США №20140357530; и публикацию патентной заявки РСТ W02014093701, включенные в настоящее описание в качестве ссылки.
Функциональные изменения и скрининг
[753] В другом аспекте осуществления настоящее изобретение относится к способу функциональной оценки и скрининга генов. Использование системы CRISPR согласно настоящему изобретению для точной доставки функциональных доменов, для активации или подавления генов или для изменения эпигенетического состояния путем точного изменения сайта метилирования в конкретном локусе-мишени может быть выполнено с использованием одной или более направляющих РНК для одной клетки, или популяции клеток, или библиотеки, примененной к геному в совокупности клеток ex vivo или in vivo, включая введение или экспрессию библиотеки, содержащей множество направляющих молекул РНК (sg-РНК), и где скрининг дополнительно включает использование эффекторного белка C2c2, причем содержащий эффекторный белок комплекс CRISPR модифицирован таким образом, чтобы содержать гетерологичный функциональный домен. В одном аспекте изобретение обеспечивает способ скрининга генома/транскриптома, включающий введение хозяину или экспрессию в хозяине библиотеки in vivo. В одном аспекте изобретение относится к способу, описанному в настоящем описании, дополнительно включающему активатор, вводимый хозяину или экспрессируемый в хозяине. В одном аспекте изобретение относится к способу, описанному в настоящем описании, в котором активатор присоединен к эффекторному белку C2c2. В одном аспекте изобретение относится к способу, описанному в настоящем описании, в котором активатор присоединен к N-концу или C-концу эффекторного белка C2c2. В одном аспекте изобретение относится к способу, описанному в настоящем описании, в котором активатор присоединен к петле sg-РНК. В одном аспекте изобретение относится к способу, описанному в настоящем описании, дополнительно включающему репрессор, вводимый хозяину или экспрессируемый в хозяине. В одном аспекте изобретение относится к способу, описанному в настоящем описании, где скрининг включает изменение и обнаружение активации гена, ингибирования гена или расщепления в конкретном локусе.
[754] В одном аспекте изобретение обеспечивает эффективную целевую активность и минимизирует нецелевую активность. В одном аспекте изобретение обеспечивает эффективное целевое расщепление эффекторным белком C2c2 и сводит к минимуму нецелевое расщепление эффекторным белком C2c2. В одном аспекте изобретение обеспечивает направляющее специфическое связывание эффекторного белка C2c2 в локусе без расщепления ДНК. Соответственно, в одном аспекте изобретение обеспечивает мишень-специфическую генную регуляцию. В одном аспекте изобретение обеспечивает направляющее специфическое связывание эффекторного белка C2c2 в локусе гена без расщепления ДНК. Соответственно, в одном варианте изобретение предусматривает расщепление в одном локусе и регуляцию гена в другом локусе с использованием одного эффекторного белка C2c2. В одном аспекте изобретение обеспечивает ортогональную активацию, и/или ингибирование, и/или расщепление множества мишеней с использованием одного или более эффекторных белков C2c2 и/или ферментов.
[755] В одном аспекте изобретение относится к способу, описанному в настоящем описании, в котором клетка-хозяин является эукариотической клеткой. В одном аспекте изобретение относится к способу, описанному в настоящем описании, в котором клетка-хозяин представляет собой клетку млекопитающего. В одном аспекте изобретение относится к способу, описанному в настоящем описании, в котором хозяин представляет собой эукариотический организм, отличный от человека. В одном аспекте изобретение относится к способу, описанному в настоящем описании, в котором эукариотический организм, отличный от человека, является млекопитающим, отличным от человека. В одном аспекте изобретение относится к способу, описанному в настоящем описании, где млекопитающее, отличное от человека, представляет собой мышь. Одним аспектом изобретения является способ, описанный в настоящем описании, включающий доставку комплексов эффекторных белков C2c2 или их компонентов или молекулы(молекул) нуклеиновой кислоты, кодирующей их, где указанная молекула(ы) нуклеиновой кислоты функционально связана(ы) с регуляторной последовательностью(ми) и экспрессируется in vivo. В одном аспекте изобретение относится к способу, описанному в настоящем описании, в котором экспрессия in vivo осуществляется с использованием лентивируса, аденовируса или AAV. В одном аспекте изобретение относится к способу, описанному в настоящем описании, в котором доставка осуществляется через частицу, наночастицу, липид или проникающий в клетку пептид (CPP).
[756] В одном аспекте изобретение относится к паре комплексов CRISPR, включающих эффекторный белок C2c2, каждый из которых содержит направляющую молекулу РНК (sg-РНК), содержащую направляющую последовательность, способную к гибридизации с последовательностью-мишенью в геномном локусе-мишени в клетке, где по меньшей мере одна петля каждой sg-РНК модифицирована вставкой отдельной последовательности(ей) РНК, которая связана с одним или более адаптерными белками, и где адаптерный белок связан с одним или более функциональными доменами, где каждая sg-РНК каждого комплекса эффекторного белка C2c2 включает функциональный домен, обладающий активностью расщепления ДНК. В одном аспекте изобретение относится к парным комплексам эффекторного белка C2c1 или C2c3, как описано в настоящем описании, где активность расщепления ДНК обусловлена нуклеазой Fok1.
[757] В одном аспекте изобретение относится к способу разрезания последовательности-мишени в представляющем интерес геномном локусе, включающему доставку к клетке комплексов эффекторного белка C2c2, или его компонента или молекулы(молекул) нуклеиновой кислоты, кодирующей их, причем молекула(ы) нуклеиновой кислоты функционально связана с регуляторной последовательностью (последовательностями) и экспрессируется in vivo. В одном аспекте изобретение относится к способу, описанному в настоящем описании, в котором доставка производится с использованием лентивируса, аденовируса или AAV. В одном аспекте изобретение относится к способу, описанному в настоящем описании, или парному комплексу эффекторного белка C2c2, как описано в настоящем описании, в котором последовательность-мишень для первого комплекса пары находится на первой цепи двухцепочечной ДНК и последовательность-мишень для второго комплекса пары находится на второй цепи двухцепочечной ДНК. В одном аспекте изобретение относится к способу, описанному в настоящем описании, или парному комплексу эффекторного белка C2c2, как описано в настоящем описании, в котором последовательности-мишени первого и второго комплексов находятся вблизи друг друга, так что ДНК разрезается таким образом, чтобы облегчить направляемую гомологией репарацию. В одном аспекте способ настоящего изобретения может дополнительно включать введение в клетки матрицы ДНК. В одном аспекте описанный в настоящем описании способ или описанные в настоящем описании парные комплексы эффекторного белка C2c2 могут вовлекать случая, где каждый комплекс эффекторного белка C2c2 имеет эффекторный фермент C2c2, в который внесена мутация таким образом, что он имеет не более чем приблизительно 5% активности нуклеазы эффекторного фермента C2c2, который не имеет мутации.
[758] В одном аспекте изобретение относится к библиотеке, способу или комплексу, как описано в настоящем описании, в которых одноцепочечная направляющая sg-РНК модифицирована, чтобы иметь по меньшей мере одну некодирующую функциональную шпильку, например, где по меньшей мере одна некодирующая функциональная шпилька является репрессивнной; например, в которой, по меньшей мере одна некодирующая функциональная петля содержит Alu.
[759] В одном аспекте изобретение относится к способу изменения или модификации экспрессии продукта гена. Указанный способ может включать введение в клетку, содержащую и экспрессирующую молекулу ДНК, кодирующую продукт гена, сконструированной, не встречающейся в природе системы CRISPR, содержащей эффекторный белок C2c2 и направляющую РНК, которая нацелена на молекулу ДНК, в результате чего направляющая РНК осуществляет нацеливание на молекулу ДНК, кодирующую продукт гена, и эффекторный белок C2c2 расщепляет молекулу ДНК, кодирующую продукт гена, в результате чего экспрессия продукта гена изменяется, и где эффекторный белок C2c2 и направляющая РНК не встречаются вместе в естественных условиях. Изобретение охватывает направляющую РНК, содержащую направляющую последовательность, связанную с последовательностью прямого повтора. Кроме того, изобретение подразумевает, что эффекторный белок C2c2 является кодон-оптимизированным для экспрессии в эукариотической клетке. В предпочтительном варианте осуществления эукариотическая клетка представляет собой клетку млекопитающего, а в более предпочтительном варианте осуществления клетка млекопитающего представляет собой клетку человека. В дальнейшем варианте осуществления изобретения экспрессия продукта гена уменьшается.
[760] В некоторых вариантах осуществления один или более функциональных доменов связаны с эффекторным белком C2c2. В некоторых вариантах осуществления один или более функциональных доменов связаны с адаптерным белком, например, с использованием модифицированных направляющих молекул Konnerman et al. (Nature 517, 583-588, 29 января 2015 года). В некоторых вариантах осуществления один или более функциональных доменов связаны с "мертвой" одноцепочечной направляющей (sg-РНК, d-РНК). В некоторых вариантах осуществления комплекс d-РНК с активным эффекторным белком C2c2 обеспечивает регуляцию гена посредством функционального домена в локусе гена, тогда как sg-РНК обеспечивает расщепление ДНК активным эффекторным белком C2c2 в другом локусе, например, как описано аналогично для систем CRISPR-Cas9 в Dahlman et al., "Orthogonal gene control with a catalytically active Cas9 nuclease" (в печати). В некоторых вариантах осуществления d-РНК выбирают так, чтобы максимизировать селективность регуляции представляющего интерес локуса гена по сравнению с неспецифической регуляцией. В некоторых вариантах осуществления d-РНК выбирают так, чтобы максимизировать регулирование гена-мишени и минимизировать расщепление мишени.
[761] Для целей последующего обсуждения "функциональный домен" может представлять собой функциональный домен, связанный с эффекторным белком C2c2, или функциональный домен, связанный с адаптерным белком.
[762] В некоторых вариантах осуществления один или более функциональных доменов представляют собой NLS (последовательность ядерной локализации) или NES (ядерный экспортный сигнал). В некоторых вариантах осуществления один или более функциональных доменов являются доменом активации транскрипции, включающим VP64, p65, MyoD1, HSF1, RTA, SET7/9 и гистонацетилтрансферу. Другие случаи упоминания в настоящем описании доменов активации (или доменов-активаторов) в отношении доменов, которые связаны с ферментом CRISPR, включают любой известный домен активации транскрипции и, в частности, VP64, p65, MyoD1, HSF1, RTA, SET7/9 или гистонацетилтрансферазу.
[763] В некоторых вариантах осуществления один или более функциональных доменов являются доменами подавления транскрипции. В некоторых вариантах осуществления доменами подавления транскрипции является KRAB-домен. В некоторых вариантах осуществления домен подавления транскрипции является доменом NuE, доменом NcoR, доменом SID или доменом SID4X.
[764] В некоторых вариантах осуществления один или более функциональных доменов имеют одну или более функциональных активностей, включающих активность активации трансляции, активность подавления трансляции, метилазную активность, деметилазную активность, активность активации транскрипции, активность подавления транскрипции, активность фактора терминации транскрипции, активность модификации гистонов, активность расщепления РНК, активность расщепления ДНК, активность интеграции ДНК или активность связывания нуклеиновых кислот.
[765] Домены модификации гистонов также предпочтительны в некоторых вариантах осуществления. Типичные домены модификации гистонов обсуждаются ниже. В качестве функциональных доменов в рамках настоящего изобретения также предпочтительными являются домены транспозазы, домены HR (гомологичной рекомбинации), домены рекомбиназы и/или интегразы. В некоторых вариантах осуществления активность интеграции ДНК включает домены HR, домены интегразы, домены рекомбиназы и/или домены транспозазы. В некоторых вариантах предпочтительны домены гистонацетилтрансферазы.
[766] В некоторых вариантах осуществления активность расщепления ДНК обусловлена нуклеазой. В некоторых вариантах осуществления нуклеаза содержит нуклеазу Fok1. См. "Dimeric CRISPR RNA-guided Fok1 nucleases for highly specific genome editing", Shengdar Q. Tsai, Nicolas Wyvekens, Cyd Khayter, Jennifer A. Foden, Vishal Thapar, Deepak Reyon, Mathew J. Goodwin, Martin J. Aryee, J. Keith Joung Nature Biotechnology 32(6): 569-77 (2014), которая относится к димерным Fok1-нуклеазам, направляемым РНК, которые распознают расширенные последовательности и могут редактировать эндогенные гены с высокой эффективностью в клетках человека.
[767] В некоторых вариантах осуществления один или более функциональных доменов присоединены к эффекторному белку C2c2, так что при связывании с одноцепочечной направляющей РНК (sg-РНК) и мишенью функциональный домен находится в пространственной ориентации, позволяющей функциональному домену выполнять свойственную ему функцию.
[768] В некоторых вариантах осуществления один или более функциональных доменов присоединены к адаптерному белку, так что после связывания эффекторного белка C2c2 с одноцепочечной направляющей РНК (sg-РНК) и мишенью функциональный домен находится в пространственной ориентации, позволяющей функциональному домену выполнять свойственную ему функцию.
[769] В одном аспекте изобретение относится к композиции, описанной в настоящем описании, в которой один или более функциональных доменов присоединены к эффекторному белку C2c2 или адаптерному белку посредством линкера, необязательно линкера GlySer, как описано в настоящем описании.
[770] Эндогенное подавление транскрипции часто опосредуется ферментами, модифицирующими хроматин, такими как гистон-метилтрансферазы (ГМТ) и деацетилазы (HDAC). Репрессивные эффекторные домены гистонов известны, и их иллюстративный список приведен ниже. В таблице предпочтение отдавалось белкам и функциональным укороченным частям небольшого размера для облегчения эффективной упаковки вирусов (например, посредством AAV). В общем, однако, домены могут включать домены деацетилазы (HDAC), гистон-метилтрансферазы (HMT) и ингибиторы гистонацетилтрансферазы (HAT), а также белки, рекрутирующие HDAC и HMT. Функциональный домен может быть или включать в некоторых вариантах осуществления эффекторные домены HDAC, рекрутирующие эффекторные домены HDAC, эффекторные домены гистонацетилтрансферазы (HMT), рекрутирующие эффекторные домены гистон-метилтрансферазы (HMT), рекрутирующие эффекторные домены гистонацетилтрансферазы.
Эффекторные домены HDAC
(Gregoretti)
H4K16Ac
H3K56Ac
(Hnisz)
(Landry)
(Wilson)
H4K16Ac
(Gertz)
(Zhu)
H3K56Ac
(Tennen)
[771] Соответственно, репрессорные домены по настоящему изобретению могут быть выбраны из гистон-метилтрансферазы (ГМТ), гистондезацетилаз (HDAC), ингибиторов гистонацетилтрансферазы (HAT), а также привлекающих HDAC и HMT белков.
[772] Домен HDAC может быть любым из приведенных в таблице выше: HDAC8, RPD3, MesoLo4, HDAC11, HDT1, SIRT3, HST2, CobB, HST2, SIRT5, Sir2A или SIRT6.
[773] В некотором варианте осуществления функциональный домен может быть рекрутирующим эффекторным доменом HDCP. Предпочтительные примеры включают те, которые приведены в таблице ниже, а именно MeCP2, MBD2b, Sin3a, NcoR, SALL1, RCOR1. NcoR проиллюстрирован в примерах настоящего описания и, хотя использование этого белка предпочтительно, предполагается, что другие представители класса также будут полезны.
Таблица HDAC рекрутирующие эффекторные домены
комплекс
(Nan)
(Boeke)
(Laherty)
взаимодействие с HDAC1
(Zhang)
(Lauberth)
(Gu, Ouyang)
[774] В некотором варианте осуществления функциональный домен может быть эффекторным доменом метилтрансферазы (HMT). Предпочтительные примеры включают те, которые приведены в таблице ниже, а именно NUE, vSET, EHMT2/G9A, SUV39H1, dim-5, KYP, SUVR4, SET4, SET1, SETD8 и TgSET8. NUE проиллюстрированы в примерах настоящего описания и, хотя использование этого белка предпочтительно, предполагается, что другие представители класса также будут полезны.
Таблица эффекторных доменов гистонметилтрансферазы (HMT)
комплекс
H3, H4
(Pennini)
SET2
G9A
H3K9,
H3K27
H1K25me1
(Tachibana)
Пре-SET
SET,
Пост-SET
(Snowden)
Пре-SET
SET,
Пост-SET
9
(Rathert)
Пре-SET
SET,
Пост-SET
(подсемейство SUVH)
(Jackson)
(подсемейство SUVR)
(Thorstensen)
Пре-SET
SET,
Пост-SET
(Vielle)
(Vielle)
(Couture)
SET
(Sautel)
SET
[775] В одном из вариантов осуществления функциональный домен может представлять собой рекрутирующий эффекторный домен гистон-метилтрансферазы (HMT). Предпочтительные примеры включают примеры, приведенные в таблице ниже, а именно Hp1a, PHF19 и NIPP1.
[776] Таблица эффективности рекрутирующих эффекторных доменов гистонметилтрансферазы (HMT).
комплекс
1
(Hathawa)
chromoshad
0
Линкер GGSG+(500-580)
( )
)
PHD2
1
(Jin)
EED
[777] В некотором варианте осуществления функциональный домен может быть ингибиторным эффекторным доменом гистонацетилтрансферазы. Предпочтительные примеры включают SET/TAF-1β, перечисленные в таблице ниже.
[778] Таблица ингибиторных эффекторных доменов гистонацетилтрансферазы.
комплекс
musculus
(Cervoni)
[779] Также является предпочтительным нацеливание на эндогенные элементы контроля (регуляторные), такие как участвующие в трансляции, придающие стабильность или, где применимо (энхансеры и сайленсеры), дополнительно к промотору или элементам, расположенным проксимально относительно промотора. Таким образом, изобретение может также использоваться для нацеливания на эндогенные элементы контроля (включая энхансеры и сайленсеры) в дополнение к нацеливанию на промотор. Эти элементы контроля могут быть расположены в восходящем- и нисходящем-направлении от точки начала транскрипции (TSS), начиная с 200 п.н. от TSS до 100 т.п.н от нее. Нацеливание на известные элементы контроля может быть использовано для активации или подавления гена-мишени. В некоторых случаях один элемент управления может влиять на транскрипцию нескольких генов-мишеней. Таким образом, нацеливания на один элемент контроля может использоваться для управления транскрипцией нескольких генов одновременно.
[780] Нацеливание на предполагаемые элементы контроля с другой стороны, (например, путем охвата области предполагаемого элемента контроля, а также от 200 п.н. до 100 т.п.н. вокруг элемента) может использоваться как средство подтверждения таких элементов (путем измерения транскрипции представляющего интерес гена) или для обнаружения новых элементов контроля (например, путем охвата области, находящейся на расстоянии 100 т.п.н. в восходящем и нисходящем направлении от TSS представляющего интерес гена). Кроме того, нацеливание на предполагаемые элементы контроля может быть полезно в контексте понимания генетических причин заболевания. Многие мутации и распространенные SNP-варианты, связанные с фенотипами заболевания, расположены за пределами кодирующих областей. После нацеливания на такие области с помощью систем активации или подавления, описанных в настоящем описании, можно провести определение данных транскрипции для либо a) набора предполагаемых мишеней (например, набора генов, расположенных в непосредственной близости от элемента контроля), либо b) для всего транскриптома, например, с помощью секвенирования РНК или микрочипа. Это позволило бы идентифицировать вероятные гены-кандидаты, вовлеченные в фенотип болезни. Такие гены-кандидаты могут быть полезны в качестве новых мишеней лекарственных препаратов.
[781] В настоящем описании упоминаются ингибиторы гистонацетилтрансферазы (HAT). Однако в некоторых вариантах осуществления альтернатива заключается в том, что один или более функциональных доменов содержат ацетилтрансферазу, предпочтительно гистонацетилтрансферазу. Они полезны в области эпигеномики, например, в способах исследования эпигенома. Способы исследования эпигенома могут включать, например, нацеливание на эпигеномные последовательности. Нацеливание на эпигемомные последовательностей может включать направляющую молекулу, направленную на эпигеномную последовательность-мишень. В некоторых вариантах осуществления эпигеномная последовательность-мишень может включать последовательность промотора, сайленсера или энхансера.
[782] Использование функционального домена, связанного с эффекторным белком C2c2, как описано в настоящем описании, предпочтительно мертвым эффекторным белком, более предпочтительно мертвым эффекторным белком FnC2c2, для нацеливания на эпигеномные последовательности возможно для активации или подавления промоторов, сайленсеров или энхансеров.
[783] Примеры ацетилтрансфераз известны, но могут включать, в некоторых вариантах осуществления, гистонацетилтрансферазы. В некоторых вариантах осуществления гистонацетилтрансфераза может содержать каталитическое ядро ацетилтрансферазы р300 человека (Gerbasch & Reddy, Nature Biotech, 6th April 2015).
[784] В некоторых предпочтительных вариантах осуществления функциональный домен связан с мертвым эффекторным белком C2c2 для нацеливания на и активации эпигеномных последовательностей, таких как промоторы или энхансеры. Одна или более направляющих молекул, нацеленных на такие промоторы или энхансеры, также могут быть предоставлены для управления связыванием фермента CRISPR с такими промоторами или энхансерами.
[785] В определенных вариантах осуществления нацеленный на РНК эффекторный белок по изобретению может использоваться для препятствования котранскрипционным модификациям структуры ДНК или хроматина, управляемому РНК метилированию ДНК или управляемому РНК сайленсингу/активации ДНК/хроматина. Управляемое РНК метилирование ДНК (RdDM) представляет собой эпигенетический процесс, впервые обнаруженный в растениях. В ходе RdDM двухцепочечные РНК (дцДНК) процессируются с образованием малых интерферирующих РНК (миРНК) длиной до 21-24 нуклеотидов и направляют метилирование гомологичных локусов ДНК. Помимо молекул РНК, множество белков участвует в создании RdDM, такие как белки Argonaute, ДНК-метилтрансферазы, комплексы ремоделирования хроматина и специфические для растений ДНК-полимераза IV и ДНК-полимераза V. Все они действуют совместно, присоединяя метильную группу в 5'-положении цитозинов. Малые РНК могут модифицировать структуру хроматина и подверженную сайленсингу транскрипцию, направляя комплексы, содержащие Argonaute, к комплементарным наращиваемым (не кодирующим) транскриптам РНК. Затем опосредуется привлечение комплексов модификации хроматина, включая гистон- и ДНК-метилтрансферазы. Нацеленный на РНК эффекторный белок по изобретению можно использовать для нацеливания на такие малые РНК и препятствования взаимодействиям между этими малыми РНК и наращиваемыми некодирующими транскриптами.
[786] Термин "ассоциированный с" используется в настоящем описании в отношении ассоциации функционального домена с эффекторным белком C2c2 или адаптерным белком. Он используется в отношении того, как одна молекула "ассоциирует" (связывается) с другой, например, между адаптерным белком и функциональным доменом, или между эффекторным белком C2c2 и функциональным доменом. В случае таких белок-белковых взаимодействий эту ассоциацию можно рассматривать с точки зрения распознавания, подобно тому, как антитело распознает эпитоп. Альтернативно, один белок может быть связан с другим белком посредством слияния двух субъединиц, например, одна субъединица является слитой с другой субъединицей. Слияние обычно происходит путем добавления одной аминокислотной последовательности к другой, например, путем совместного сплайсинга нуклеотидных последовательностей, которые кодируют каждый белок или субъединицу. Альтернативно, это может по существу рассматриваться как связывание двух молекул или прямое образование связи, такое как слитый белок. В любом случае слитый белок может включать линкер между представляющими интерес субъединицами (то есть между ферментом и функциональным доменом или между адаптерным белком и функциональным доменом). Таким образом, в некоторых вариантах осуществления эффекторный белок C2c2 или адаптерный белок ассоциирован с функциональным доменом путем связывания с ним. В других вариантах осуществления эффекторный белок C2c2 или адаптерный белок ассоциирован с функциональным доменом, потому что оба слиты вместе, необязательно посредством промежуточного линкера.
Насыщающий мутагенез
[787] Система(ы) эффекторного белка C2c2, описанная в настоящем описании, может быть использована для выполнения насыщающего или глубоко сканирующего мутагенеза геномных локусов в сочетании с клеточным фенотипом, например, для определения критических минимальных признаков и дискретных уязвимостей функциональных элементов, необходимых для экспрессии генов, лекарственной устойчивости и купирования заболевания. Под "насыщающим" или "глубоко сканирующим мутагенезом" подразумевается, что каждое или по существу каждое основание ДНК разрезается внутри геномных локусов. В популяцию клеток можно ввести библиотеку направляющих РНК эффекторных белков C2c2. Библиотека может быть введена таким образом, что каждая клетка получает одну направляющую молекулу РНК (sg-РНК). В случае, когда библиотека вводится путем трансдукции вирусного вектора, как описано в настоящем описании, используется низкая множественность инфекции. Библиотека может включать sg-РНК, нацеленные на каждую последовательность в восходящем направлении от последовательности, прилегающей к протоспейсеру (PAM) в геномном локусе. Библиотека может включать по меньшей мере 100 непересекающихся геномных последовательностей в восходящем направлении от PAM для каждых 1000 пар оснований в геномном локусе. Библиотека может включать sg-РНК, нацеливающие на последовательности в восходящем направлении по меньшей мере от одной отличающейся PAM. Системы эффекторного белка C2c2 могут включать более одного белка C2c2. Можно использовать любой эффекторный белок C2c2, как описано в настоящем описании, включая ортологи или сконструированные эффекторные белки C2c2, которые распознают различные последовательности PAM. Частота не являющихся мишенями сайтов для sg-РНК может составлять менее 500. Оценка нецелевого связывания sg-РНК необходима для отбора sg-РНК с наименьшим уровнем возможного связывания с не являющимися мишенями участками. Любой фенотип, который, как установлено, связан с разрезанием участке-мишени sg-РНК, может быть подтвержден с использованием sg-РНК, нацеленных на один и тот же сайт в одном эксперименте. Валидация участка-мишени также может быть выполнена с использованием модифицированного эффекторного белка C2c2, как описано в настоящем описании, и двух sg-РНК, нацеливающих на участок-мишень в геноме. Без связи с теорией, можно утверждать, что участок-мишень действительно установлен, если изменение фенотипа наблюдается в экспериментах по проверке достоверности.
[788] Что касается описанных в настоящем описании белков, нацеленных ДНК, геномные локусы могут включать по меньшей мере одну непрерывную геномную область. По меньшей мере одна непрерывная геномная область может включать и весь геном. По меньшей мере одна непрерывная геномная область может содержать функциональный элемент генома. Функциональный элемент может находиться в некодирующей области, кодирующем гене, интронной области, промоторе или энхансере. По меньшей мере одна непрерывная геномная область может содержать по меньшей мере 1 т.п.н., предпочтительно по меньшей мере 50 т.п.н. геномной ДНК. По меньшей мере одна непрерывная геномная область может содержать сайт связывания фактора транскрипции. По меньшей мере одна непрерывная геномная область может содержать область, гиперчувствительную к расщеплению ДНК-азой I. По меньшей мере одна непрерывная геномная область может содержать усиливающий транскрипцию энхансер или подавляющий транскрипцию репрессорный элемент. По меньшей мере одна непрерывная геномная область может содержать сайт, обогащенный эпигенетической сигнатурой. По меньшей мере одна непрерывная область геномной ДНК может содержать эпигенетический инсулятор. По меньшей мере, одна непрерывная геномная область может содержать две или более непрерывных геномных областей, которые физически взаимодействуют. Геномные области, которые взаимодействуют, могут определяться "технологией 4C". 4C-технология позволяет провести скрининг всего генома непредвзятым образом для сегментов ДНК, которые физически взаимодействуют с выбранным фрагментом ДНК, как описано в Zhao et al. ((2006) Nat Genet 38, 1341-7) и в патенте США 8642295, которые включены в настоящее описание в качестве ссылок в полном объеме. Эпигенетическая сигнатура может представлять собой ацетилирование, метилирование, убиквитинацию, фосфорилирование гистонов, метилирование ДНК или отсутствие всего вышеперечисленного.
[789] Система(ы) эффекторного белка C2c2 для насыщающего или глубоко сканирующего мутагенеза может быть использована в популяции клеток. Система эффекторного белка C2c2 может использоваться в эукариотических клетках, включая, но не ограничиваясь ими, клетки млекопитающих и растений. Популяция клеток может состоять из прокариотических клеток. Популяция эукариотических клеток может быть популяцией эмбриональных стволовых (ES) клеток, нейронов, эпителиальных клеток, иммунных клеток, эндокринных клеток, мышечных клеток, эритроцитов, лимфоцитов, растительных клеток или дрожжевых клеток.
[790] В одном аспекте настоящее изобретение относится к способу скрининга функциональных элементов, связанных с изменением фенотипа. Библиотека может быть введена в популяцию клеток, которые адаптированы для содержания эффекторного белка C2c2. Клетки могут быть рассортированы по меньшей мере на две основные группы на основе фенотипа. Фенотип может включать экспрессию гена, рост или жизнеспособность клеток. Может быть определена относительная представленность направляющих РНК, присутствующих в каждой группе, причем геномные сайты, связанные с изменением фенотипа, определяются по представленности направляющих РНК, присутствующих в каждой группе. Изменение фенотипа может быть изменением экспрессии гена-мишени. Экспрессия гена-мишени может быть повышена, понижена, или полностью выключена (нокаут гена). Клетки могут быть отсортированы в группы с высокой и низкой экспрессией. Популяция клеток может включать репортерную конструкцию, которая определяет фенотип. Конструкция репортера может включать поддающийся обнаружению маркер. Клетки могут быть отсортированы с помощью поддающегося обнаружению маркера.
[791] В другом аспекте настоящее изобретение относится к способу скрининга геномных сайтов, связанных с устойчивостью к химическому соединению. Химическое соединение может быть лекарственным средством или пестицидом. Библиотека может быть введена в популяцию клеток, адаптированных к содержанию эффекторного белка C2c2, где каждая клетка популяции содержит не более одной направляющей РНК; популяцию клеток обрабатывают химическим соединением; и представленность направляющих РНК определяют после обработки химическим соединением в более поздний момент времени по сравнению с ранним моментом времени, при котором геномные участки, связанные с устойчивостью к химическому соединению, определяются по обогащению направляющими РНК. Представленность sg-РНК может быть определена способами глубокого секвенирования.
[792] Полезными в практике настоящего изобретения с использованием комплексов эффекторных белков C2c2 являются способы, в которых используются системы CRISPR-Cas9, и может быть приведена ссылка на статью под названием "BCL11A enhancer dissection by Cas9-mediated in situ saturating mutagenesis". Canver, M.C., Smith,E.C., Sher, F., Pinello, L., Sanjana, N.E., Shalem, O., Chen, D.D., Schupp, P.G., Vinjamur, D.S., Garcia, S.P., Luc, S., Kurita, R., Nakamura, Y., Fujiwara, Y., Maeda, T., Yuan, G., Zhang, F., Grkin, S.H., & Bauer, D.E. DOI: 10.1038/naturel552I, published online September 16, 2015, которая включена в настоящее описание в качестве ссылки и кратно обсуждается ниже.
[793] Canver et al. использует новые объединенные библиотеки направляющих cr-РНК-Cas9, чтобы выполнять насыщающий мутагенез in situ человеческих и мышиных эритроидных энхансеров BCL11A, ранее идентифицированных как энхансер, ассоциированный с уровнем фетального гемоглобина (HbF), и мышиный ортолог которого необходим для экспрессии эритроида BCL11A. Этот подход выявил критические минимальные признаки и дискретную уязвимость этих энхансеров. Благодаря редактированию первичных человеческих предшественников и трансгенеза мышей, авторы подтвердили, что эритроидный энхансер BCL11A может быть использован в качестве мишени для реиндукции HbF. Авторы создали подробную карту энхансера, которая может быть использована при терапевтическом редактировании генома.
Способы использования систем C2c2 для модификации клетки или организма
[794] В некоторых вариантах осуществления изобретение относится к способу модификации клетки или организма. Клетка может быть прокариотической или эукариотической. Клетка может быть клеткой млекопитающего. Клетка может быть не принадлежащей человеку клеткой млекопитающего, клеткой примата, коровы, свиньи, грызуна или мыши. Клетка может быть эукариотической клеткой не млекопитающего, такой как клетка домашней птицы, рыбы или креветки. Клетка также может быть растительной клеткой. Растительная клетка может быть клеткой зерновой культуры, такой как маниока, кукуруза, сорго, пшеница или рис. Растительная клетка может быть клеткой водоросли, дерева или овоща. Модификация, введенная в клетку согласно настоящему изобретению, может быть такой, что клетка и потомки клетки изменяются для улучшения производства биологических продуктов, таких как антитела, крахмал, спирт или другой желаемый клеточной продукции. Модификация, введенная в клетку согласно настоящему изобретению, может быть такой, что клетка и ее потомки включают изменение, которое изменяет продуцируемый ими биологический продукт.
[795] Описываемая система может содержать один или более различных векторов. В одном аспекте изобретения эффекторный белок является кодон-оптимизированным для экспрессии в желаемом типе клеток, предпочтительно эукариотических клеток, предпочтительно клеток млекопитающего или клеток человека.
[796] Упаковочные клетки обычно используются для получения вирусных частиц, которые способны инфицировать клетку-хозяина. Такие клетки включают клетки 293, которые упаковывают аденовирусы и клетки ψ2 или клетки РА317, которые упаковывают ретровирусы. Вирусные векторы, используемые в генной терапии, обычно получают путем создания клеточной линии, которая производит упаковку векторов нуклеиновой кислоты в вирусные частицы. Векторы обычно содержат минимальные вирусные последовательности, необходимые для упаковки и последующей интеграции в клетку-хозяина, при этом другие вирусные последовательности заменяются кассетой экспрессии для полинуклеотида(ов), подлежащего(их) экспрессии. Отсутствующие функции вируса обычно поставляются в процессе доставки упаковочными клетками линии. Например, векторы AAV, используемые в генной терапии, обычно имеют только последовательности ITR из генома AAV, которые необходимы для упаковки и интеграции в геном хозяина. Вирусная ДНК упаковывается с помощью клеточной линии, которая содержит вспомогательную плазмиду, кодирующую другие гены AAV, а именно rep и cap, но не содержащую последовательность ITR. Линия клеток также может быть инфицирована аденовирусом в качестве вспомогательной частицы. Вспомогательный вирус способствует репликации вектора AAV и экспрессии генов AAV из вспомогательной плазмиды. Вспомогательная плазмида не может быть упакована в больших количествах из-за отсутствия последовательности ITR. Контаминация аденовирусом может быть уменьшена, например, с помощью термообработки, к которой аденовирус более чувствителен, чем AAV. Квалифицированным специалистам в данной области известны дополнительные способы доставки нуклеиновых кислот в клетки. См., например, US 20030087817, включенную в настоящее описание в качестве ссылки.
[797] В некоторых вариантах осуществления клетку-хозяина временно или постоянно трансфицируют одним или более векторами, описанными в настоящем описании. В некоторых вариантах осуществления клетку трансфицируют в том виде, в каком она присутствует у индивидуума естественным образом. В некоторых вариантах осуществления изобретения клетку, которую трансфицируют, извлекают из субъекта, такого как клеточная линия. В данной области известно большое разнообразие клеточных линий для тканевой культуры. Примеры клеточных линий включают, но не ограничиваются ими: C8161, CCRF-CEM, MOLT, mIMCD-3, NHDF, HeLa-S3, Huh1, Huh4, Huh7, HUVEC, HASMC, HEKn, HEKa, MiaPaCell, Panc1, PC-3, TF1, CTLL-2, C1R, Rat6, CV1, RPTE, A10, T24, J82, A375, ARH-77, Calu1, SW480, SW620, SKOV3, SK-UT, CaCo2, P388D1, SEM-K2, WEHI-231, HB56, TIB55, Jurkat, J45.01, LRMB, Bcl-1, ВС-3, IC21, DLD2, Raw264.7, NRK, NRK-52E, MRC5, MEF, Hep G2, HeLa B, HeLa T4, COS, COS-1, COS-6, COS-M6A, эпителий почки обезьяны BS-C-1, фибробласты эмбрионов мыши BALB/3T3, 3T3 Swiss, 3T3-L1, 132-d5 фетальные фибробласты человека, фибробласты мыши 10.1, 293-Т, 3T3, 721, 9L, A2780, A2780ADR, A2780cis, A172, A20, A253, A431, A-549, ALC, B16, B35, BCP-1, BEAS-2B, bEnd.3, BHK-21, BR 293, ВхРСЗ, C3H-10T1/2, C6/36, Cal-27, CHO, CHO-7, CHO-IR, CHO-K1, CHO-K2, CHO-T, CHO Dhfr -/-, COR-L23, COR-L23/CPR, COR-L23/5010, COR-L23/R23, COS-7, COV-434, CML T1, CMT, CT26, D17, DH82, DU145, DuCaP, EL4, EM2, EM3, EMT6/AR1, EMT6/AR10.0, FM3, H1299, H69, HB54, HB55, HCA2, HEK-293, HeLa, Hepa1c1c7, HL-60, HMEC, HT-29, Jurkat, JY клетки, клетки K562, Ku812, KCL22, KG1, KYO1, LNCap, Ma-Mel 1-48, MC-38, MCF-7, MCF-10A, MDA-MB-231, MDA-MB-468, MDA-MB-435, MDCK II, MDCK II, MOR/0.2R. MONO-MAC 6, MTD-1A, MyEnd, NCI-H69/CPR, NCI-H69/LX10, NCI-H69/LX20, NCI-H69/LX4, NIH-3T3, NALM-1, NW-145, OPCN /OPCT клеточные линии, Peer, PNT-1A/PNT 2, RenCa, RIN-5F, RMA/RMAS, клетки Saos-2, Sf-9, SkBr3, T2, T-47D, T84, THP1, U373, U87, U937, VCaP, клетки Vero, WM39, WT-49, X63, YAC-1, YAR и их трансгенные разновидности. Клеточные линии доступны из множества источников, известных квалифицированным специалистам в данной области (см., например, Американскую коллекцию типовых культур (ATCC) (Manassus, Va.)). В некоторых вариантах осуществления клетка, трансфицированная одним или более векторами, описанными в настоящем описании, используется для получения новой клеточной линии, содержащей одну или более последовательностей, полученных из вектора. В некоторых вариантах осуществления клетка, временная трансфицированная компонентами системы, нацеленной на нуклеиновую кислоту, как описано в настоящем описании (например, путем временной трансфекции одного или более векторов или трансфекции РНК) и модифицированная посредством активности комплекса нацеливания на нуклеиновую кислоту, используется для создания новой клеточной линии, содержащей клетки, имеющие модификацию, но не имеющие какой-либо другой экзогенной последовательности. В некоторых вариантах осуществления изобретения клетки, временно или постоянно трансфицированные одним или более векторами, описанными в настоящем описании, или клеточные линии, полученные из таких клеток, используются для оценки одного или более тестируемых соединений.
[798] В некоторых вариантах осуществления изобретения один или более векторов, описанных в настоящем описании, используются для получения трансгенного животного, отличного от человека, или трансгенного растения. В некоторых вариантах осуществления трансгенное животное представляет собой млекопитающее, такое как мышь, крыса или кролик. В некоторых вариантах осуществления организм или субъект является растением. В некоторых вариантах осуществления организм, субъект или растение являются водорослями. Способы получения трансгенных растений и животных известны в данной области и обычно начинаются с использования способа трансфекции клеток, как описано в настоящем описании.
[799] В одном аспекте изобретение относится к способам модификации полинуклеотида-мишени в эукариотической клетке. В некоторых вариантах осуществления такой способ включает позволение комплекса, нацеленного на нуклеиновую кислоту, связываться с полинуклеотидом-мишенью для расщепления указанного полинуклеотида-мишени, тем самым модифицируя полинуклеотид-мишень, причем комплекс, нацеленный на нуклеиновую кислоту, включает эффекторный белок, нацеленный на нуклеиновую кислоту, и направляющую РНК, гибридизованную с последовательностью-мишенью в указанном полинуклеотиде-мишени.
[800] В одном аспекте изобретение относится к способу модификации экспрессии полинуклеотида в эукариотической клетке. В некоторых вариантах осуществления изобретения способ включает позволение комплексу, нацеленному на нуклеиновую кислоту, связываться с полинуклеотидом, так что указанное связывание приводит к увеличению или уменьшению экспрессии указанного полинуклеотида; где комплекс, нацеленный на нуклеиновую кислоту, содержит эффекторный белок, нацеленный на нуклеиновую кислоту, связанный с направляющей РНК, которая гибридизована с последовательностью-мишенью в указанном полинуклеотиде.
Комплексы эффекторного белка C2c2 могут использоваться в растениях
[801] Система(ы) эффекторного белка C2c2 (например, одиночная или мультиплексированная) может использоваться в сочетании с новейшими достижениями в геномике культур. Описанную в настоящем описании систему можно использовать для выполнения результативного и экономически эффективного исследования или манипуляции с геном или геномом растений, например, для быстрого исследования, и/или отбора, и/или выбора, и/или сравнения, и/или манипуляций, и/или трансформации растительных генов или геномов, например, для создания, идентификации, разработки, оптимизации или присвоения определенной характеристики(характеристик) или признака (набора признаков) растениям или для трансформации генома растения. Таким образом, может быть улучшено производство растений, а именно новых растений с новыми комбинациями признаков или характеристик или новых растений с улучшенными признаками. Система или системы из эффекторного белка C2c2 может использоваться на растениях при использовании следующих способов: сайт-направленной интеграции (SDI) или редактирования генов (GE), близкого обратного скрещивания (NRB) или обратного скрещивание (RB). Аспекты использования описанных в настоящем описании эффекторных систем C2c2 могут быть аналогичны использованию систем CRISPR-Cas (например, CRISPR-Cas9) в растениях. В этой связи заслуживает упоминания веб-сайт Университета Аризоны "CRISPR-PLANT" (http:/www.genome.arizona.edu/crispr/) (поддерживается PennState и AGI). Варианты осуществления изобретения могут быть использованы при редактировании генома в растениях или в тех случаях, где ранее применялись способы с использованием иРНК или аналогичных способов редактирования генома; см., например, Nekrasov, "Plant genome editing made easy: targeted mutagenesis in model and crop plants using the CRISPR-Cas system," Plant Methods 2013, 9:39 (doi: 10.1186/1746-4811-9-39); Brooks, "Efficient gene editing in tomato in the first generation using the CRISPR-Cas9 system," Plant Physiology September 2014 pp 114.247577; Shan, "Targeted genome modification of crop plants using a CRISPR-Cas system," Nature Biotechnology 31, 686-688 (2013); Feng, "Efficient genome editing in plants using a CRISPR/Cas system," Cell Research (2013) 23:1229-1232 doi: 10.1038/cr.2013.114; published online 20 August 2013; Xie, "RNA-guided genome editing in plants using a CRISPR-Cas system," Mol Plant. 2013 Nov; 6(6):1975- 83. doi: 10.1093/mp/sstl 19. Epub 2013 Aug 17; Xu, "Gene targeting using the Agrobacterium tumefaciens-mediated CRISPR-Cas system in rice," Rice 2014, 7:5 (2014), Zhou et af., "Exploiting SNPs for biallelic CRISPR mutations in the outcrossing woody perennial Populus reveals 4-coumarate: CoA ligase specificity and Redundancy," New Phytologist (2015) (Forum) 1-4 (доступен через интернет по ссылке wmv.newphytologist.com); Caliando et al, "Targeted DNA degradation using a CRISPR device stably carried in the host genome, NATURE COMMUNICATIONS 6:6989, DOI: 10.1038/ncomms7989, vvwvv.nature.com/naturecommunications DOI: 10.1038/ncomms7989; патент США №6603061 - Agrobacterium-Mediated Plant Transformation Method; патент США №7868149 - Plant Genome Sequences and Uses Thereof, и US 2009/0100536 - Transgenic Plants with Enhanced Agronomic Traits, содержание каждого из которых включено в настоящее описание в качестве ссылки в полном объеме. В практическом воплощении изобретения, содержание и раскрытие Morrell et al. "Crop genomics: advances and applications", NatRevGenet. 2011 Dec 29; 13 (21:85-96; включено в настоящее описание в качестве ссылки, в том числе в отношении того, как данное изобретение может быть применено к растениям. Соответственно, данная ссылка может также применяться и к клеткам животных, с учетом возможных различий, к растительным клеткам, если не указано иное; и в этом случае ферменты, обладающие сниженными неспецифическими эффектами и системы, использующие такие ферменты, могут быть использованы применительно к растениям, в том числе к тем, которые упомянуты в настоящей заявке.
[802] Sugano et al. (Plant Cell Physiol. 2014 Mar; 55(3):475-81. doi: 10.1093/pcp/pcu014. Epub 2014 Jan 18) сообщает о применении CRISPR-Cas9 для направленного мутагенеза печеночника Marchantiapolymorpha L., который стал использоваться как модель для экспериментов для изучения эволюции высших растений. Промотор U6 M. polymorpha идентифицировали и клонировали для экспрессии гРНК. Последовательность, являющаяся мишенью гРНК, была разработана для нарушения работы гена, кодирующего фактор ауксина 1 (ARF1) у M. polymorpha. Используя трансформацию при помощи Agrobacterium, Sugano et al. получили устойчивые мутанты в фазе гаметофита для M. polymorpha. При использовании CRISPR-Cas9 сайт-направленный мутагенез in vivo был получен с помощью промотора мозаики цветной капусты 35S или промотора M. polymorphaEF1α для экспрессии Cas9. Изолированные мутантные индивидуумы, демонстрирующие ауксин-устойчивый фенотип, не были химерами. Более того, стабильные мутанты были получены путем бесполого размножения растений T1. Многочисленные аллели arf1 легко получали с использованием направленного мутагенеза на основе системы CRIPSR/Cas9. Системы C2c2 согласно настоящему изобретению могут быть использованы для регулирования тех же, а также других генов, и сходных систем контроля экспрессии, таких как РНК-i и миРНК. Способ по изобретению может быть индуцируемым и обратимым.
[803] Kabadi et al. (Nucleic Acids Res. 2014 Oct 29; 42(19):e147. doi: 10.1093/nar/gku749. Epub 2014 Aug 13) разработали единую лентивирусную систему, чтобы экспрессировать вариант Cas9, ген-репортер и до четырех sg-РНК с независимых промоторов РНК-полимеразы III, которые включены в вектор с помощью достаточно удобного способа клонирования GoldenGate. Каждая sg-РНК была эффективно экспрессирована и может опосредовать редактирование мультиплексного гена и устойчивую активацию транскрипции в иммортализованных и первичных клетках человека. Настоящее изобретение может быть использовано для регулирования генов растений Kabadi.
[804] Xing et al. (BMCPlantBiology 2014, 14: 327) разработали бинарный векторный набор CRISPR-Cas9 на основе каркаса pGreen или pCAMBIA, а также гРНК. Этот набор инструментов не требует каких-либо дополнительных ферментов рестрикции помимо BsaI для создания конечных конструкций с высокой эффективностью всего за один шаг клонирования. Эти конструкции могут содержать кодон-оптимизированный для кукурузы Cas9 и одну или более гРНК. Инструментарий был подтвержден с использованием протопластов кукурузы, трансгенных линий кукурузы и трансгенных линий Arabidopsis и, как было показано, демонстрирует высокую эффективность и специфичность воздействия. Однако, что видится еще более важным, с использованием этого инструментария были обнаружены направленные мутации трех генов Arabidopsis в трансгенных ростках поколения T1. Более того, мутации с множественными генами могут быть унаследованы следующим поколением. Набор векторных модулей (направляющая РНК) использовался в качестве инструментария для мультиплексного редактирования генома в растениях. Системы C2c2 и белки согласно настоящему изобретению могут использоваться для нацеливания на гены-мишени согласно Xing.
[805] Системы CRISPR C2c2 по изобретению могут использоваться для обнаружения вирусов растений. Gambino et al. (Phytopathology. 2006 Nov; 96(11): 1223-9. doi: 10.1094/PHYTO-96-1223) полагались на амплификацию и мультиплексную ПЦР для одновременного обнаружения девяти вирусов виноградной лозы. Аналогичным образом системы C2c2 и белки по настоящему изобретению могут быть использованы для обнаружения множества мишеней в растении-хозяине. Кроме того, системы по изобретению могут быть использованы для одновременного нокдауна экспрессии вирусного гена в ценных агрикультурах и предотвращения активации или дальнейшего заражения путем нацеливания на экспрессию вирусной РНК.
[806] Murray et al. (ProcBiolSci., 2013 Jun 26, 280 (1765): 20130965. doi: 10.1098/rspb.2013.0965, опубликовано 22 августа 2013 года) проанализировали 12 растительных РНК-вирусов, чтобы выявить скорость эволюции, и обнаружили доказательства эпизодического отбора, возможно, происходившего из-за сдвигов между различными генотипами или видами хозяина. Системы C2c2 и белки согласно настоящему изобретению могут быть использованы для нацеливания на или иммунизации против таких вирусов в хозяине. Например, системы по изобретению могут быть использованы для выключения экспрессии вирусной РНК, а следовательно, и репликации. Кроме того, изобретение может быть использовано как для нацеливания нуклеиновых кислот на расщепления, так и для нацеливания на экспрессию или активацию. Кроме того, системы по изобретению могут быть мультиплексированы таким образом, чтобы поражать несколько целей или множественный изолят того же вируса.
[807] Ma et al. (Mol Plant, 2015 Aug 3, 8 (8): 1274-84. Doi: 10.1016./j.molp.2015.04.007) сообщают о надежной векторной системе CRISPR-Cas9, использующей кодон-оптимизированный для растений ген Cas9 для удобного и высокоэффективного мультиплексного редактирования генома в однодольных и двудольных растениях. Ma et al. разработали специальные процедуры на основе ПЦР, для быстрого получения множественных экспрессирующих кассет для sg-РНК, которые могут быть собраны в бинарные векторы CRISPR-Cas9 за один раунд клонирования путем лигирования по типу GoldenGate или сборки Гиббса (GibsonAssembly). В этой системе Ma et al. отредактировали 46 целевых сайтов риса со средней скоростью мутации 85,4%, в основном в биаллельном и гомозиготном состоянии. Ma et al. приводят примеры моделирования генной мутации - потери функции в растениях риса T0 и Arabidopsis T1 путем одновременного нацеливания на несколько (до восьми) членов семейства генов, множественно генов в биосинтетическом пути или множество участков в одном гене. Аналогично, системы C2c2 по настоящему изобретению могут одновременно и успешно быть нацелены на экспрессию нескольких генов.
[808] Lowder et al. (PlantPhysiol. 2015 Aug 21. doi: pp.00636.2015) также разработали набор инструментов CRISPR-Cas9, позволяющий редактировать мультиплексный геном и транскрипционную регуляцию экспрессированных генов, генов с подавленной экспрессией или некодирующих генов в растениях. Этот набор инструментов предоставляет исследователям протокол и реагенты для быстрой и эффективной сборки функциональных конструкций T-ДНК CRISPR-Cas9 для однодольных и двудольных растений с использованием способов клонирования GoldenGate и Gateway. Он предоставляется с полным набором возможных инструментов, включая мультиплексированное редактирование генов и активацию транскрипции или репрессирование эндогенных генов растений, технологию трансформации на основе Т-ДНК, и имеет фундаментальное значение для современной биотехнологии растений, генетики, молекулярной биологии и физиологии. Поэтому, авторы изобретения разработали способ сборки Cas9 (WT, никазы или dCas9) и одной или более гРНК в представляющий интерес конечный Т-ДНК-вектор. Способ сборки основан как на типе сборки GoldenGate, так и на рекомбинации MultiSite Gateway. Для сборки требуются три модуля. Первый модуль представляет собой исходный вектор Cas9, который содержит гены Cas9 без промоторов или производные от него гены, фланкированные сайтами attL1 и attR5. Второй модуль представляет собой вектор записи гРНК, который содержит входную гРНК. Экспрессирующие кассеты фланкированы сайтами attL5 и attL2. Третий модуль включает attR1-attR2-содержащие конечные Т-ДНК-векторы, которые обеспечивают промоторы выбора для экспрессии Cas9. Инструментарий Lowder et al. могут быть применен к системе эффекторного белка C2c2 согласно настоящему изобретению.
[809] Организмы, такие как дрожжи и микроводоросли, широко используются в синтетической биологии. Stovicek et al. (Metab.Eng. Comm., 2015; 2:13) описывают редактирование генома промышленных дрожжей, например Saccharomyces cerevisae, для эффективного создания устойчивых штаммов в целях промышленного производства. Stovicek использовал кодон-оптимизированную систему CRISPR-Cas9 для дрожжей, чтобы одновременно производить как разрыв аллели эндогенного гена, так и нокин гетерологичного гена. Cas9 и гРНК экспрессировали в геномных или эписомальных положениях на векторе 2μ. Авторы также показали, что эффективность разрушения гена может быть улучшена путем оптимизации уровней экспрессии Cas9 и гРНК. Hlavova et al. (Biotechnol, Adv., 2015) описали развитие видов или штаммов микроводорослей с использованием таких способов, как CRISPR, для нацеливания на ядерные и хлоропластные гены в целях инсерционного мутагенеза и скрининга. Те же самые плазмиды и векторы могут быть применены к системам C2c2 по настоящему изобретению.
[810] Petersen ("Towards precisely glycol engineered plants,", Plant Biotech Denmark Annual Meeting 2015, Копенгаген, Дания) разработал способ использования CRISPR/Cas9 для разработки изменений генома в Arabidopsis, например, для глико-преобразования Arabidopsis в целях производства белков и продуктов, имеющих желаемые посттрансляционные модификации. Hebeistrup et al. (FrontPlantSci., 2015, апрель 23, 6:247) излагают принципы подобных изменений для биоинженерии крахмала непосредственно в растениях, создавая культуры, которые экспрессируют ферменты, модифицирующие крахмал, и непосредственно синтезируют продукты, которые обычно производятся промышленными химическими и/или физическими способами обработки крахмалов. Способы, предложенные Petersen и Hebeistrup, могут быть применены и к эффекторной белковой системе C2c2 согласно настоящему изобретению.
[811] Kurthe et al., J Virol. 2012 Jun, 86 (11): 6002-9. Doi: 10.1128/JVI.00436-12. Epub 2012 Mar 21) разработали вектор на основе РНК-вируса для внедрения желаемых признаков в виноградную лозу без наследственных модификаций генома. Вектор обеспечивал способность регулировать экспрессию эндогенных генов путем подавления экспрессии гена при помощи вируса. Системы и белки C2c2 по настоящему изобретению могут использоваться для подавления экспрессии генов и белков без наследуемой модификации генома.
[812] В одном варианте осуществления изобретения растение может быть бобовым. Настоящее изобретение может использовать описанную в настоящем описании систему CRISP-Cas для изучения и модификации, в качестве неограничивающего примера, соевых бобов, гороха и арахиса. Curtin et al. предоставляет набор инструментов для функциональной геномики бобовых. (См. Curtin et al., "A genome engineering toolbox for legume Functional genomics", International Plant and Animal Genome Conference XXII 2014). Curtin использовал генетическую трансформацию CRISPR для нокаута/нокдауна одиночной копии и дублированных генов бобовых как в культуре "волосатых" корней, так и в целых системах растений. Некоторые из генов-мишеней были выбраны для изучения и оптимизации особенностей систем нокаут/нокдаун (например, фитоэндесатуразы), в то время как другие были идентифицированы по гомологии генов сои с Dicer-подобным генами Arabidopsis или с помощью исследований полногеномной ассоциации для нодуляции в Medicago. Системы и белки C2c2 по настоящему изобретению могут быть применены в системах нокаута/нокдауна.
[813] Аллергические реакции на арахис и бобовые обычно являются серьезной проблемой для здоровья. Система на основе эффекторного белка C2c2 согласно настоящему изобретению может быть использована для идентификации, а затем и редактирования или подавления экспрессии генов, кодирующих аллергенные белки таких бобовых. Без ограничений касательно таких генов и белков Nicolaou et al. идентифицировали аллергенные белки в арахисе, соевых бобах, чечевице, горохе, люпине, зеленой фасоли и маше (бобах мунг). См. Nicolaou et al., Current Opinion in Allergy and Clinical Immunology 2011; 11 (3): 222).
[814] В предпочтительном варианте осуществления изобретения растение может быть деревом. Настоящее изобретение также может использовать описанную в настоящем описании систему CRISPR-Cas для трав (см., например, Belhaj et al., Plant Methods 9: 39 и Harrison et al., Genes&Development, 28: 1859-1872). В особенно предпочтительном варианте осуществления система CRISPR-Cas по настоящему изобретению может быть нацелена на однонуклеотидный полиморфизм (SNP) в деревьях (см., например, Zhou et al., NewPhytologist, Volume 208, Issue 2, pages 298-301, October 2015). Zhou et al. применили систему CRISPR-Cas на древесном многолетнем Populus с использованием семейства генов 4-кумарата:КоА-лигазы (4CL) в качестве примера и достигли 100% мутационной эффективности для двух преобразованных таким образом генов 4CL, при этом каждый трансформированный организм был исследован на предмет переноса двуаллельных модификаций. В исследовании Zhou et al. система CRISPR-Cas9 была очень чувствительна к однонуклеотидным полиморфизмам (SNP), поскольку расщепление третьего гена 4CL устранялось из-за SNP в последовательности-мишени. Эти способы могут быть применены к системе эффекторного белка C2c2 согласно настоящему изобретению.
[815] Способы Zhou et al. (NewPhytologist, Volume 208, Issue 2, pages 298-301, October 2015) можно применить к настоящему изобретению следующим образом. Два гена 4CL, 4CL1 и 4CL2, связанные с биосинтезом лигнина и флавоноидов, соответственно, являются мишенью для редактирования CRISPR-Cas9. Populustremula x alba клон 717-1B4, обычно используемый для трансформации, отличается от генотипа Populus trichocarpa. Таким образом, гРНК 4CL1 и 4CL2, сконструированные на основе эталонного генома, могут быть проанализированы при помощи собственно полученных данных 717 секвенирований РНК, чтобы убедиться в отсутствии SNP, которые могут ограничивать эффективность Cas. Также предусматривается третья гРНК, предназначенная для 4CL5, дубликата 4CL1. Соответствующие 717 последовательностей содержат один SNP в каждом аллеле вблизи/внутри PAM, оба из которых, как ожидается, устраняют нацеливание посредством 4CL5-гРНК. Все три сайта-мишени гРНК расположены в пределах первого экзона. Для 717 трансформаций гРНК экспрессируется с промотора Medicago U6.6 вместе с кодон-оптимизированным Cas человека под контролем промотора CaMV 35S в бинарном векторе. Трансформация только с помощью вектора Cas может служить в качестве контроля. Случайно выбранные линии 4CL1 и 4CL2 подвергаются секвенированию ампликонов. Затем данные обрабатываются, а биаллельные мутации подтверждаются во всех случаях. Эти способы могут быть применены к системе эффекторного белка C2c2 согласно настоящему изобретению.
[816] В растениях патогены часто специфичны по отношению к хозяину. Например, Fusariumoxysporum f. sp. lycopersici поражает только томат, а F. oxysporum f. dianthii Pucciniagraminis f. sp. tritici только пшеницу. Растения имеют естественную и индуцируемую защиту, чтобы противостоять большинству патогенов. Мутации и явления рекомбинации между поколениями растений приводят к генетической изменчивости, которая приводит к восприимчивости к болезням, особенно в том случае, когда патогены воспроизводятся с большей частотой, чем растения. В растениях может наблюдаться "нехозяйская" устойчивость, например, когда хозяин и патоген не совместимы. Также может наблюдаться горизонтальная устойчивость, например, частичная устойчивость к аллелям расы патогенных организмов, обычно контролируемая многими генами, и вертикальная устойчивость, например, полная устойчивость к некоторым расам патогена, но не к другим расам, обычно контролируемым несколькими генами. На уровне отношений "ген против гена" растения и патогены развиваются вместе, происходит извечное нарушение и восстановление баланса: генетические изменения в одном приводят к нарушениям в другом. Соответственно, используя природную вариативность генома, селекционеры объединяют в одном растении наиболее полезные гены для урожайности, качества, однородности, выносливости, сопротивления различным болезням. Источниками генов устойчивости являются местные или иностранные сорта, старые сорта негибридного происхождения, родственники диких растений и сорта с индуцированной мутацией, например, после обработки растительного материала мутагенными агентами. Так, в соответствии с настоящим изобретением селекционерам предлагается новый инструмент стимуляции мутаций. Соответственно, специалист в данной области сможет проанализировать геномы разных сортов - возможных источников резистентных генов, имеющих желаемые характеристики или признаки, и сможет использовать настоящее изобретение, чтобы индуцировать появление генов устойчивости со значительно большей точностью, чем предыдущие мутагенные агенты, что, следовательно, приведет к ускорению и улучшению селекции растений.
[817] Помимо растений, которые обсуждались здесь и выше, модифицированные способами инженерии растения включают также растения, измененные эффекторным белком и соответствующей направляющей молекулой, а также их потомство, если таковое предусмотрено. Они могут включать культуры, устойчивые к болезням или засухе, такие как пшеница, ячмень, рис, соя или кукуруза; растения, модифицированные для полного подавления или уменьшения способности к самоопылению (но которые, по возможности, способны к образованию гибридов); и аллергенные продукты питания, такие как арахис и орехи, где иммуногенные белки были отключены, разрушены или разрушены путем нацеливания через эффекторный белок и подходящую направляющую молекулу.
Терапевтическое лечение
[818] Система по изобретению может быть применена в областях ранее применявшихся технологий производства разрыва РНК без проведения излишних экспериментов. Исходя из настоящего описания, возможные применения включают терапевтические, аналитические и другие прикладные задачи, поскольку настоящая заявка обеспечивает основу для осознанного конструирования способами инженерии системы. Настоящее изобретение относится к терапевтическому лечению заболевания, вызванного сверхэкспрессией РНК, токсичной РНК и/или мутантной РНК (такой как, имеющей дефекты сплайсинга или усечение). Экспрессия токсичной РНК может быть связана с образованием ядерных включений и поздними дегенеративными изменениями в мозге, сердце или скелетной мышце. В наиболее изученном примере болезни Штейнерта или миотонической дистрофии оказывается, что основным патогенным результатом токсической РНК является секвестрирование связываемых белков и нарушение регуляции альтернативного сплайсинга (Hum.Mol. Genet. (2006) 15 (suppl 2): R162-R169). Миотоническая дистрофия [dystrophia myotonica (DM)] интересна генетикам, поскольку она демонстрирует чрезвычайно широкий спектр клинических признаков, неполное перечисление которых может включает мышечное истощение, катаракту, резистентность к инсулину, атрофию яичка, замедление сердечной проводимости, кожные опухоли и влияние на когнитивные функции. Классическая форма DM, которая теперь называется DM 1 типа (DM1), вызвана распространением CTG-повторов в 3'-нетранслируемой области (UTR) DMPK, гена, кодирующего цитозольную протеинкиназу.
[819] В приведенной ниже таблице представлен список экзонов, для которых были показаны нарушения в альтернативном сплайсинге в скелетоной (поперечно-полосатой) мышечной ткани, сердце или головном мозге при болезни первого типа - DM1.
Скелетная (поперечно-полосатая) мышечная ткань

Сердце


Головной мозг

[820] Ферменты по настоящему изобретению могут быть нацелены на сверхэкспрессированную РНК или токсичную РНК, например, такую как РНК гена DMPK или любого неправильно регулируемого альтернативного сплайсинга при заболевании DM1 в скелетной мышце, сердце или головном мозге, которые приведены, например, в таблице выше.
[821] Ферменты по настоящему изобретению также могут быть нацелены на транс-действующие мутации, влияющие на РНК-зависимые функции, которые вызывают заболевание (обобщенно представлены в Cell., 2009, февраль 20, 136 (4): 777-793), как указано в таблице ниже.
Дискератоз врожденный (сцепленный с Х-хромосомой/ аутосомно-рецессивный)
[822] Фермент по настоящему изобретению также может быть использован для лечения различных таупатий, включая первичные и вторичные таупатии, такие как первичная возрастная таупатия (PART)/старческое слабоумие с преобладанием нейрофибриллярных клубков, нейрофибриллярные клубки (NFT), сходные с таковыми при Болезни Альцгеймера (AD), но без образования бляшек, dementia pugilistica (хроническая травматическая энцефалопатия), прогрессирующий супрануклеарный парез взора, кортикобазальная дегенерация, лобно-височная деменция и паркинсонизм, связанные с хромосомой 17, болезнь Айото-Бодига (комплекс Паркинсона-деменции Гуама), ганглиоглиома и ганглиоцитома, менингиоангиоматоз, постэнцефалитный паркинсонизм, подострый склерозирующий панэнцефалит, а также свинцовая энцефалопатия, туберозный склероз, болезнь Халвервордена-Спатца и липофусциноз, болезнь Альцгеймера. Ферменты по настоящему изобретению также могут быть нацелены на мутации, нарушающие цис-действующий код сплайсинга, вызывают дефекты сплайсинга и заболевание (обзор в Cell, 2009 Feb 20; 136 (4): 777-793). Болезнь двигательного нейрона (SMA) связана с делецией гена SMN1. Оставшийся ген SMN2 имеет C->T-замещение в экзоне 7, которое инактивирует экзонный энхансер сплайсинга (ESE) и создает экзонный сайленсер сплайсера (ESS), что приводит к тому, что экзон 7 пропускается и получается усеченный белок (SMNA7). Замена T-> A в экзоне 31 гена дистрофина одновременно создает кодон преждевременной терминации (STOP) и экзонный сайленсер сплайсинга (ESS), приводящий к пропусканию экзона 31. Эта мутация вызывает умеренную форму DMD, потому что мРНК, лишенная экзона 31, продуцирует частично функциональный белок. Мутации внутри и после экзона 10 гена МАРТ, кодирующего белок tau, влияют на регуляторные элементы сплайсинга и нарушают нормальное соотношение мРНК 1:1, включая или исключая экзон 10. Это приводит к нарушенному равновесию между белками tau, содержащими либо четыре, либо три микротрубочки (4R-tau и 3R-tau соответственно), вызывая невропатологическое расстройство FTDP-17. Приведенный пример представляет собой мутацию N279K, которая усиливает функцию ESE, способствующую включению экзона 10, и смещение баланса в сторону увеличения 4R-tau. Полиморфные (UG)m(U)n тракты в 3'- сайте сплайсинга в экзоне 9 гена CFTR влияют на степень включения экзона 9 и уровень полноразмерного функционального белка, варьируя тяжесть муковисцидоза (CF), вызванного мутациями в другом месте в гене CFTR.
[823] Врожденная иммунная система обнаруживает вирусную инфекцию прежде всего путем распознавания вирусных нуклеиновых кислот внутри инфицированной клетки, обозначаемой ДНК или РНК-чувствительностью. Анализы чувствительности РНК in vitro могут быть использованы для обнаружения специфических субстратов РНК. Нацеленный на РНК эффекторный белок может быть, например, использован для обнаружения РНК в живых клетках. Примером таких применений является диагностика путем выявления специфичных для болезни РНК.
[824] Эффекторный белок, нацеленный на РНК, согласно изобретению может также быть использован для противовирусной активности, в частности против вирусов РНК. Эффекторный белок может быть нацелен вирусную РНК при использовании подходящей направляющей РНК, избирательной для выбранной последовательности вирусной РНК. В частности, эффекторный белок может быть активной нуклеазой, которая расщепляет РНК, например одноцепочечную. Таким образом, предлагается использование нацеленного на РНК эффекторного белка в качестве противовирусного агента согласно изобретению.
[825] Терапевтические дозы ферментной системы согласно настоящему изобретению для нацеливания на вышеописанные типы РНК предполагаются в дозировках, равных от примерно 0,1 до примерно 2 мг/кг, которые могут вводиться последовательно с контролем ответа и при необходимости повторным дозированием до 7-10 доз на пациента. Преимущественно образцы получают от каждого пациента во время проведения лечения для определения эффективности лечения. Например, образцы РНК могут быть выделены и количественно определены, чтобы определить, уменьшена или увеличена экспрессия. Проведение диагностики такого рода находится в компетенции специалистов в данной области.
[826] Что касается наиболее общей информации по системам CRISPR-Cas, то заявители упоминают следующие работы (также включенные в настоящую заявку посредством отсылки)
- Multiplex genome engineering using CRISPR/Cas systems. Cong, L, Ran, F.A., Cox, D., Lin, S., Barretto, R., Habib, N., Hsu, PI), Wu, X., Jiang, W., Marraffmi, L.A., & Zhang, F. Science Feb 15; 339(6121):819-23 (2013);
- RNA-guided editing of bacterial genomes using CRISPR-Cassystems. Jiang W., Bikard D., Cox D., Zhang F, Marraffmi LA. Nat Biotechnol Mar; 31(3):233-9 (2013);
- One-Step Generation of Mice Carrying Mutations in Multiple Genes by CRISPR/Cas - Mediated Genome Engineering. Wang H., Yang R, Shivalila CS., Dawlaty MM., Cheng AW., Zhang F., Jaeniseh R. Cell May 9; 153(4); 910-8 (2013);
- Optical control of mammalian endogenous transcription and epigenetic states. Konermann S, Brigham AID, Trevino AE, Hsu PD, Heidenreich M, Cong L, Platt RJ, Scott DA, Church GM, Zhang F. Nature. Aug 22;500(7463):472-6. doi; 10.1038/Naturel2466. Epub 2013 Aug 23 (2013);
- Double Nicking by RNA-Guided CRISPR Cas9 for Enhanced Genome Editing Specificity. Ran, FA., Hsu, PD., Lin, CY., Gootenberg, JS., Konermann, S., Trevino, AE., Scott, DA \, Inoue, A., Matoba, S., Zhang, Y., & Zhang, F. Cell Aug 28, pii: S0092-8674(13)01015-5 (2013-A);
- DNA targeting specificity of RNA-guided Cas9 nucleases, Hsu, P., Scott, D, Weinstein, J., Ran, FA., Konermann, S., Agarwala, V., Li, Y., Fine, E., Wu, X, Shalem, O, Cradick, TJ, Marraffini, LA, Bao, G., & Zhang, F. Nat Biotechnol doi:10.1038/nbt.2647 (2013);
- Genome engineering using the CRISPR-Cas9 system. Ran, FA, Hsu, PD., Wright J., Agarwala, V, Scott, DA., Zhang, F, Nature Protocols Nov; 8(11):2281-308 (2013-B);
- A Genome-Scale CRISPR-Cas9 Knockout Screening in Human Cells, Shalem, Q., Sanjana, NE., Hartenian, E., Shi, X., Scott, DA., Mikkelson, T., Heckl, D., Ebert, BE., Root, DE, Doench, JG, Zhang, F. Science Dec 12. (2013). [Epub ahead of print];
- Crystal staicture of cas9 in complex with guide RNA and target DNA. Nishimasu, H., Ran, FA., Hsu, PD., Konermann, S., Shehata, SI, Dohmae, N., Ishitani, R., Zhang, F., Nureki, (), Cell Feb 27, 156(5):935-49 (2014);
- Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells. Wu X., Scott DA., Kriz AJ., Chiu AC., Hsu PD., Dadon DB, Cheng AW., Trevino AE, Konermann S., Chen S., Jaenisch R., Zhang F., Sharp PA. Nat Bioteehnol. Apr 20. doi: 10. lQ38/nbt.2889 (2014);
- CRISPR-Cas9 Knockin Mice for Genome Editing and Cancer Modeling. Platt RJ, Chen S, Zhou Y, Yim MJ, Swiech L, Kempton HR, Dahlman JE, Parnas G, Eisenhaure TM, Jovanovic M, Graham DB, Jhunjhunwala S, Heidenreich M, Xavier RJ, Danger R, Anderson DG, Hacohen N, Regev A, Feng G, Sharp PA, Zhang F. Cell 159(2): 440-455 DOI: 10.1016/j. cell.2014.09.014 (2014);
- Development and Applications of CRISPR-Cas9 for Genome Engineering, Hsu PD, Lander ES, Zhang F., Cell. Jun 5, 157(6): 1262-78 (2014).
- Genetic screens in human cells using the CRISPR/Cas9 system, Wang T, Wei JJ, Sabatini DM, Lander ES., Science. January 3; 343(6166): 80-84. doi: 10.1126/science. 1246981 (2014);
- Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation, Doench JG, Hartenian E, Graham DB, Tothova Z, Hegde M, Smith I, Sullender M, Ebert BL, Xavier RJ, Root DE., (published online 3 September 2014) Nat Bioteehnol. Dec;32(12): 1262-7 (2014);
- In vivo interrogation of gene function in the mammalian brain using CRISPR-Cas9, Swiech L, Heidenreich M, Banerjee A, Habib N, Li Y, Trombetta J, Sur M, Zhang F., (published online 19 October 2014) Nat Bioteehnol, Jan, 33(1): 102-6 (2015);
- Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex, Konermann S, Brigham MD, Trevino AE, Joung J, Abudayyeh OO, Barcena C, Hsu PD, Habib N, Gootenberg JS, Nishimasu H, Nureki O, Zhang F., Nature. Jan 29; 517(7536):583-8 (2015).
- A split-Cas9 architecture for inducible genome editing and transcription modulation, Zetsche B, Volz SE, Zhang F., (published online 02 February 2015) Nat Bioteehnol. Feb; 33(2): 139-42 (2015);
- Genome-wide CRISPR Screen in a Mouse Model of Tumor Growth and Metastasis, Chen S, Sanjana NE, Zheng K, Shalem O, Lee K, Shi X, Scott DA, Song J, Pan JQ, Weissleder R, Lee H, Zhang F, Sharp PA. Cell 160, 1246-1260, March 12, 2015 (multiplex screen in mouse), and
- In vivo genome editing using Staphylococcus aureus Cas9, Ran FA, Cong L, Yan WX, Scott DA, Gootenberg JS, Kriz AJ, Zetsche B, Shalem Q, Wu X, Makarova KS, Koonin EV, Sharp PA, Zhang F., (published online 01 April 2015), Nature. Apr 9;520(7546): 186-91 (2015).
- Shalem et. al, "High-throughput functional genomics using CRISPR-Cas9," Nature Reviews Genetics 16, 299-311 (May 2015).
- Xu et al., "Sequence determinants of improved CRISPR sgRNA design," Genome Research 25, 1147-1157 (August 2015).
- Parnas et al., "A Genome-wide CRISPR Screen in Primary Immune Cells to Dissect Regulatory Networks," Cell 162, 675-686 (July 30, 2015).
- Ramanan et al., CRISPR/Cas9 cleavage of viral DNA efficiently suppresses hepatitis В virus," Scientific Reports 5:10833, doi: 10.1038/srepl0833 (June 2, 2015),
- Nishimasu et al., Crystal Structure of Staphylococcus aureus Cas9," Cell 162, 1113-1126 (Aug, 27, 2015),
- BCL11A enhancer dissection by Cas9-mediated in situ saturating mutagenesis, Canver et al., Nature 527(7577): 192-7 (Nov. 12, 2015) doi: 10.1038/naturel5521. Epub 2015 Sep 16.
- Cpf1 Is a Single RNA-Guided Endonuclease of a Class 2 CRISPR-Cas System, Zetsche et al..Cell 163, 759-71 (Sep 25, 2015),
- Discovery and Functional Characterization of Diverse Class 2 CRISPR-Cas Systems.Shmakov et al, Molecular Cell, 60(3), 385-397 doi: 10.1016/j.molcel.2015,10.008 Epub October 22, 2015.
- Rationally engineered Cash nucleases with improved specificity, Slaymaker et al., Science 2016 Jan 1, 351(6268): 84-88 doi: 10.1126/science.aad5227. Epub 2015 Dec 1. [Epub ahead of print],
каждый из которых включен в настоящее описание в качестве ссылки, может рассматриваться в практике настоящего изобретения и кратко обсуждается ниже:
- Cong et al. разработали системы CRISPR-Cas типа II для использования в эукариотических клетках на основе как Streptococcus thermophilus Cas9, так и Streptococcus pyogenes Cas9 и показали, что нуклеазы Cas9 могут быть направлены короткой РНК, чтобы индуцировать точное расщепление ДНК в клетках человека и мыши. Их исследование также показало, что Cas9, превращенный в создающий однонитевые разрывы фермент, может быть использован для облегчения гомологической репарации в эукариотических клетках с минимальной мутагенной активностью. Кроме того, их исследование показало, что несколько направляющих последовательностей могут быть закодированы в единую последовательность CRISPR, чтобы обеспечить одновременное редактирование нескольких участков на эндогенных геномных локусах в геноме млекопитающих, демонстрируя легкость в программировании и широкую применимость технологии нуклеазы, основанной на РНК. Эта возможность использовать РНК для программирования последовательность-специфичного расщепления ДНК в клетке определила новый класс инструментов для геномной инженерии. Эти исследования также показали, что другие локусы CRISPR, вероятно, могут быть трансплантированы в клетки млекопитающих для опосредования расщепления в целевых участках генома млекопитающих. Важно отметить, что в будущем некоторые аспекты системы CRISPR-Cas могут быть дополнительно улучшены для повышения ее эффективности и универсальности.
- Jiang et al. использовали короткие палиндромные повторы, регулярно расположенные кластерами (CRISPR), связанные с эндонуклеазой Cas9, объединенные с двойными РНК, для введения точных мутаций в геномы Streptococcus pneumoniae и Escherichia coli. Этот подход основан на двойной РНК: расщепление, направляемое Cas9, на целевом геномном участке уничтожает немутированные клетки и позволяет обойти необходимость выбора маркеров или систем отрицательного отбора. В исследовании сообщается о перепрограммировании специфики комплекса двойной РНК: Cas9 путем изменения последовательности короткой CRISPRPHK (cr-РНК) для внесения одно- и мультинуклеотидные изменений в матрицы редактирования. Исследование показало, что одновременное использование двух cr-РНК обеспечивало мультиплексный мутагенез. Кроме того, когда подход использовался в сочетании с рекомбинацией, у S. pneumoniae почти 100% клеток, которые были восстановлены с использованием описанного подхода, содержали желаемую мутацию, а в E.coli 65% клеток, которые были восстановлены, содержали мутацию.
- Wang et al., 2013 использовали систему CRISPR/Cas для одношаговой генерации мышей, несущих мутации в нескольких генах, которые традиционно генерировались на нескольких стадиях путем последовательной рекомбинации в эмбриональных стволовых клетках и/или трудоемким скрещиванием мышей с единственная мутация. Система CRISPR/Cas значительно ускорит исследование in vivo функционально избыточных генов и эпистатических генных взаимодействий.
- Konermann et al. (2013) рассмотрели необходимость в универсальных и надежных технологий для оптической и химической модуляции ДНК-связывающих доменов на основе фермента CRISPR Cas9, а также подобных эффекторам активаторов транскрипции (TALE).
- Ran et al. (2013-А) описали подход, в котором объединены никазный мутант Cas9 с парными направляющими РНК для введения целенаправленных двунитевых разрывов. Это касается нуклеазы Cas9 из микробной системы CRISPR-Cas, которая нацелена на конкретные геномные локусы с помощью направляющей последовательности, которая может переносить определенные несоответствия с ДНК-мишенью и тем самым способствовать нежелательному нецелевому мутагенезу. Поскольку отдельные однонитевые разрывы в геноме восстанавливаются с высокой точностью, одновременное внесение однонитевых разрывов с помощью надлежащим образом смещенных направляющих РНК требуется для двухцепочечных разрывов и увеличивает количество специфично распознаваемых оснований для целевого расщепления. Авторы продемонстрировали, что использование парного внесения одноцепочечных разрывов может снизить нецелевую активность в 50-100 раз в клеточных линиях и облегчить нокаут генов у зигот мышей без ущерба для эффективности расщепления мишеней. Эта универсальная стратегия позволяет использовать широкий спектр приложений для редактирования генома, которые требуют высокой специфичности.
- Hsu et al. (2013) охарактеризовали специфичность нацеливания, осуществляемого SpCas9 в клетках человека, чтобы получить данные о выборе целевых сайтов и избегать нецелевого воздействия. В исследовании оценивали> 700 вариантов направляющей РНК и индуцированные SpCas9 уровни инсерции/делеции (indel-мутации)на > 100 предсказанных геномных нецелевых локусов в клетках 293T и 293FT. Авторы показали, что SpCas9 допускают несоответствия между направляющей РНК и ДНК-мишенью в разных положениях зависимым от последовательности образом, чувствительны к числу, положению и распределению несоответствий. Авторы также показали, что SpAas9-опосредованное расщепление не подвержено влиянию метилирования ДНК и что доза SpCas9 и одноцепочечной направляющей РНК (sgRNA) может быть титрована для минимизации нецелевой модификации. Кроме того, для облегчения применения приложений для инженерии генома млекопитающих авторы разработали программного обеспечения для выбора и проверки целевых последовательностей, а также нецелевого анализа.
- Ran et al. (2013-B) описали набор инструментов для Cas9-опосредованного редактирования генома путем негомологичного соединения концов (NHEJ) или гомологичной репарации (HDR) в клетках млекопитающих, а также создания модифицированных клеточных линий для последующих функциональных исследований. Для минимизации нецелевого расщепления, авторы описали стратегию внесения двойных одноцепочечных разрывов, использующую никазный мутант Cas9 с парными направляющими РНК. Протокол, предоставленный авторами, экспериментально выработал руководящие принципы для выбора целевых участков, оценки эффективности расщепления и анализа нецелевой активности. Исследования показали, что, начиная с разработки мишени, модификации генов могут быть достигнуты всего за 1-2 недели, а модифицированные клональные клеточные линии могут быть получены в течение 2-3 недель.
- Shalem et al. описали новый способ исследования функции гена в масштабе всего генома. Их исследования показали, что доставка полногеномной библиотеки нокаутов CRISPR-Cas9 (GeCKO), нацеленной на 18080 генов с 64751 уникальными направляющими последовательностями, позволила провести скрининг c помощью как отрицательного, так и положительного отбора в клетках человека. Во-первых, авторы показали использование библиотеки GeCKO для идентификации генов, необходимых для жизнеспособности клеток при раке и плюрипотентных стволовых клеток. Далее, в модели меланомы авторы провели скрининг для поиска генов, потеря которых связана с резистентностью к вемурафенибу, терапевтическому препарату, который ингибирует мутантную протеинкиназу BRAF. Их исследования показали, что кандидаты высшего ранга включали ранее проверенные гены NF1 и MED12, а также новые: NF2, CUL3, TADA2B и TADA1. Авторы наблюдали высокий уровень согласованности между независимыми направляющими РНК, нацеленными на один и тот же ген, и высоким показателем подтверждения полученного результата, и, таким образом, продемонстрировали возможность скрининга целого генома с помощью Cas9.
- Nishimasu et al. исследовали кристаллической структуре Cas9 Streptococcus pyogenes в комплексе с одноцепочечной направляющей РНК (sg-РНК) и его ДНК-мишени при разрешении 2,5 A°. Обнаружена "двулопастная" архитектура структуры, состоящая из субъединицы для распознавания мишени и нуклеазной субъединицы, вмещающих гетеродуплекс sg-РНК:ДНК в положительно заряженном желобке на их поверхности. В то время как субъединица ("доля") для распознавания необходима для связывания sg-РНК и ДНК, нуклеазная субъединица ("доля") содержит домены HNH и RuvC-нуклеазы, которые должным образом расположены для расщепления комплементарных и не комплементарных цепей ДНК-мишени соответственно. Нуклеазная субъединица также содержит карбоксильный концевой домен, ответственный за взаимодействие с последовательностью, прилегающей к протоспейсеру (PAM). Эта структура с высоким разрешением и сопутствующие функциональные анализы выявили молекулярный механизм РНК-направленного нацеливания на ДНК, осуществляемого Cas9, таким образом прокладывая путь для рационального проектирования новых, универсальных технологий редактирования генома.
- Wu et al. картировали во всем геноме сайты связывания каталитически неактивного Cas9 (dCas9) из Streptococcus pyogenes, нагруженных одноцепочечными направляющими РНК (sg-РНК) в эмбриональных стволовых клетках мыши (mESC). Авторы показали, что каждая из четырех sg-РНК тестировала мишени dCas9 между десятками и тысячами геномных участков, часто характеризующихся 5-нуклеотидной областью-затравкой в sg-РНК и NGG-соседним мотивом, прилегающим к протоспейсеру (PAM). Недоступность хроматина уменьшает связывание dCas9 с другими сайтами с соответствующими "seed"-последовательностями; таким образом, 70% нецелевых сайтов связаны с генами. Авторы показали, что целенаправленное секвенирование 295 сайтов связывания dCas9 в mESC, трансфицированных каталитически активным Cas9, выявило только один сайт, мутированный выше фоновых уровней. Авторы предложили модель с двумя состояниями для связывания и расщепления Cas9, в которой совпадение "seed"-последовательности вызывает связывание, но для расщепления требуется обширное спаривание оснований с ДНК-мишенью.
- Platt et al. создали Cre-зависимую мышь с нокином Cas9. Авторы продемонстрировали как in vivo, так и ex vivo редактирование генома с использованием аденоассоциированного вируса (AAV), лентивируса или опосредованной частицами доставки направляющей РНК в нейроны, иммунные клетки и эндотелиальные клетки.
- Hsu et al. (2014) - обзорная статья, в которой обсуждается история CRISPR-Cas9 от йогурта до редактирования генома, включая генетический скрининг клеток.
- Wang et al. (2014) относится к объединенному способу генетического скрининга, основанному на потере функциональности, подходящему как для положительного, так и для отрицательного отбора, который использует полногеномную лентивирусную библиотеку одноцепочечной направляющей РНК (sg-РНК).
- Doench et al. создали пул одноцепочечных направляющих РНК (sg-РНК), чередуя все возможные целевые сайты группы из шести эндогенных мышиных и трех эндогенных генов человека и количественно оценили их способность продуцировать нулевые аллели их целевого гена путем окрашивания антителом и проточной цитометрии. Авторы показали, что оптимизация PAM улучшила активность, а также предоставила интерактивный инструмент для разработки одноцепочечных направляющих sg-РНК.
- Swiech et al. продемонстрировали, что AAV-опосредованное редактирование генома с помощью SpCas9 может позволить провести ретроспективные генетические исследования функции гена в головном мозге.
- Konermann et al. (2015) обсуждают возможность присоединения нескольких эффекторных доменов, например активаторов транскрипции, функциональных и эпигеномных регуляторов в соответствующих положениях на направляющей молекуле, таких как шпилька или тетрапетля с линкерами и без них.
- Zetsche et al. демонстрируют, что фермент Cas9 можно разделить на два и, следовательно, можно контролировать сборку Cas9 для активации.
- Chen et al. c помощью мультиплексного полногеномного скрининга in vivo посредством CRISPR-Cas9 обнаружили у мышей гены, регулирующие метастазы в легкие.
- Ran et al. (2015) исследовали SaCas9 и его способность редактировать геномы и продемонстрировали, что нельзя экстраполировать биохимические анализы. Shalem et al. (2015) описаны способы использования каталитически неактивные слияния Cas9 (dCas9) для синтетического подавления (CRISPRi) или активации (CRISPRa) экспрессии и продемонстрированы преимущества использования Cas9 для полногеномного скрининга, включая упорядоченный и объединенные скрининг, нокаутные подходы, инактивирующие геномные локусы, и стратегии, модулирующие активность транскрипции.
Окончательные изменения
- Shalem et al. (2015) описали способы использования каталитически неактивные слияния Cas9 (dCas9) для синтетического подавления (CRISPRi) или активации (CRISPRa) экспрессии и продемонстрированы преимущества использования Cas9 для полногеномного скрининга, включая упорядоченный и объединенные скрининг, нокаутные подходы, инактивирующие геномные локусы, и стратегии, модулирующие активность транскрипции.
- Xu et al. (2015) оценили характеристики последовательности ДНК, которые способствуют эффективности одноцепочечной направляющей РНК (sg-РНК) в скрининге на основе CRISPR. Авторы исследовали эффективность нокаута CRISPR/Cas9 и предпочтение нуклеотидов на участке расщепления. Авторы также обнаружили, что предпочтительные последовательности для CRISPR-интерференции/CRISPR-активации (CRISPRi/a) существенно отличаются от таковых для нокаута CRISPR/Cas9.
- Paraas et al. (2015) ввели полногеномные объединенные библиотеки CRISPR-Cas9 в дендритные клетки для идентификации генов, контролирующих индукцию фактора некроза опухолей (Tnf) бактериальным липополисахаридом (LPS). Известные регуляторы сигналинга Tlr4 и ранее неизвестные кандидаты были идентифицированы и классифицированы на три функциональных модуля с явным влиянием на канонические ответы на LPS.
- Ramananetal (2015) продемонстрировали расщепление вирусной эписомальной ДНК (cccDNA) в инфицированных клетках. Геном HBV существует в ядрах инфицированных гепатоцитов в виде двухцепочечных эписомальных ДНК длиной 3,2 т.п.н., которые называются ковалентно замкнутой кольцевой ДНК (cccDNA), которая является ключевым компонентом жизненного цикла HBV, репликация которого не ингибируется современной терапией. Авторы показали, что одноцепочечные направляющие молекулы РНК (sg-РНК), специально предназначенные для высококонсервативных областей HBV, эффективно подавляют репликацию вируса и обедненную cccDNA.
- Nishimasu et al. (2015) сообщили о кристаллических структурах SaCas9 в комплексе с одной направляющей РНК (sg-РНК) и ее двухцепочечными ДНК-мишенями, содержащими 5'-TTGAAT-3' PAM-последовательность и 5'-TTGGGT-3' PAM-последовательность. Структурное сравнение SaCas9 с SpCas9 выявило как консервативность структуры, так и расхождение, объяснив отчетливую специфичность их PAM и ортологическое распознавание sg-РНК.
- Canver et al. (2015) продемонстрировали функциональное исследование не кодирующих геномных элементов на основе CRISPR-Cas9. Авторы разработали объединенные библиотеки направляющей РНК CRISPR-Cas9 для насыщающего мутагенеза insitu энхансеров BCL11A человека и мыши, которые выявили важные особенности этих энхансеров.
- Zetsche et al. (2015) сообщили о характеристике Cpf1, нуклеазы класса 2 CRISPR из Francisellanovicida U112, имеющей особенности, отличные от Cas9. Cpf1 представляет собой единственную РНК-направляемую эндонуклеазу, не имеющую tracr-РНК, использующую T-богатый мотив, прилегающий к протоспейсеру, и расщепляющую ДНК путем ступенчатого двухцепочечного разрыва.
- Shmakov et al. (2015) сообщили о трех разных классах систем CRISPR-Cas. Два фермента системы CRISPR (C2c1 и C2c3) содержат RuvC-подобные эндонуклеазные домены, отдаленно связанные с Cpf1. В отличие от Cpf1, C2c1 зависит как от cr-РНК, так и от tracr-РНК для расщепления ДНК. Третий фермент (C2c2) содержит два предсказанных РНК-азных домена HEPN и является независимым от tracr-РНК.
- Slaymaker et al. (2016) сообщили об использовании структурной белковой инженерии для улучшения специфичности Cas9 Streptococcus pyogenes (SpCas9). Авторы разработали варианты "расширенной специфичности" SpCas9 (eSpCas9), которые поддерживали устойчивое целевое расщепление с уменьшением нецелевых эффектов.
[827] Кроме того, "Dimeric CRISPR RNA-guided Fok1 nucleases for highly specific genome editing", Shengdar Q. Tsai, Nicolas Wyvekens, CydKhayter, Jennifer A. Foden, Vishal Thapar, Deepak Reyon, Mathew J, Goodwin, Martin J, Aryee, J. Keith Joung Nature Biotechnology 32(6): 569-77 (2014), относится к димерным РНК-направленным Fok1-нуклеазам, которые распознают расширенные последовательности и могут редактировать эндогенные гены с высокой эффективностью в клетках человека.
[828] Что касается общей информации о системах CRISPR-Cas, их компонентах и доставке таких компонентов, включаяс пособы, материалы, транспортные средства доставки, векторы, частицы, AAV, а также их изготовление и использование, в том числе что касается количества и формул, все полезное на практике для настоящего изобретения, приводится в настоящем описании в качестве ссылки на следующие номера патентов США: №8999641, 8993233, 8945839, 8932814, 8906616, 8895308, 8889418, 8889356, 8871445, 8865406, 8795965, 8771945 и 8697359; публикация заявки на патент США US 2014-0310830 (US APP.Ser. No. 14/105031), US 2014-0287938 A1 (US App. Ser. No. 14/213991), US 2014-0273234 A1 (U.S. App, Ser. No. 14/293674), US 2014-0273232 A1 (U.S. App. Ser. No. 14/290575), US 2014-0273231 (U.S. App. Ser. No. 14/259420), US 2014-0256046 A1 (U.S. App. Ser. No. 14/226274), US 2014-0248702 A1 (U.S. App. Ser. No. 14/258458), US 2014-0242700 A1 (U.S. App. Ser. No. 14/222930), US 2014-0242699 A1 (U.S. App. Ser. No, 14/183512), US 2014-0242664 A1 (U.S. App. Ser. No. 14/104990), US 2014-0234972 A1 (U.S, App. Ser. No. 14/183471), US 2014-0227787 A1 (U.S. App. Ser. No. 14/256912), US 2014-0189896 A1 (U.S. App. Ser. No. 14/105035), US 2014-0186958 (U.S. App. Ser. No. 14/105017), US 2014-0186919 Al (U.S. App. Ser. No. 14/104977), US 2014-0186843 A1 (U.S. App. Ser. No. 14/104900), US 2014-0179770 A1 (US. App. Ser. No. 14/104837) и US 2014-0179006 A1 (US. App. Ser. No. 14/183,486), US 2014-0170753 (US App. Ser. No. 14/183429), US 2015-0184139 (US. App. Ser. No. 14/324960), 14/054414; Европейские патенты: EP 2 764 103 (EP 13824232.6), EP 2 784 162 (EP 14170383.5) и EP 2 771 468 (EP 13818570.7); и публикации патентных заявок PCT: WO 2014/093661 (PCT/US 2013/074743), WO 2014/093694 (PCT/US 2013/074790), WO 2014/093595 (PCT/US 2013/074611), WO 2014/093718 (PCTAJS 2013/074825), WO 2014/093709 (PCT/US 2013/074812), WO 2014/093622 (PCT/US 2013/074667), WO 2014/093635 (PCT/US 2013/074691), WO 2014/093655 (PCT/US 2013/07473 6), WO 2014/093712 (PCT/US 2013/074819), WO 2014/093701 (PCT/US 2013/074800), WO 2014/018423 (PCT/US 2013/051418), WO 2014/204723 (PCT/US 2014/041790), WO 2014/204724 (PCT/US 2014/041800), WO 2014/204725 (PCTAJS 2014/041803), WO 2014/204726 (PCT.AJS 2014/041804), WO 2014/204727 (PCT/US 2014/041806), WO 2014/204728 (PCT/US 2014/041808), WO 2014/204729 (PCT/US 2014/041809), WO 2015/089351 (PCT/US 2014/069897), WO 2015/089354 (PCT/US 2014/069902), WO 2015/089364 (PCT/US 2014/069925), WO 2015/089427 (PCT7US 2014/070068), WO 2015/089462 (PCT/US 2014/070127), WO 2015/089419 (PCT/US 2014/070057), WO 2015/089465 (PCT/US 2014/070135), WO 2015/089486 (PCT/US 2014/070175), PCT/US 2015/051691, PCT/US 2015/051830. Также упоминается предварительная патентная заявка США 61/758468; 61/802174; 61/806375; 61/814263; 61/819803 и 61/828130, поданная 30 января 2013 года; 15 марта 2013 года; 28 марта 2013 года; 20 апреля 2013 года; 6 мая 2013 года и 28 мая 2013 года соответственно. Также упоминается предварительная патентная заявка США 61/836123, поданная 17 июня 2013 года. См. Также дополнительные заявки на патент США 61/835931, 61/835936, 61/836127, 61/836101, 61/836080 и 61/835973, поданные 17 июня 2013 года. Дальнейшая ссылка делается на временные заявки на патент США 61/862468 и 61/862355, поданные 5 августа 2013 года, 61/871301, поданная 28 августа 2013 года, 61/960777, поданную 25 сентября 2013 года и 61/961980, поданную 28 октября 2013 года. Далее делается ссылка на: PCX/US 2014/041803, PCT/US 2014/041809, PCT/US 2014/041804 и PCT/US 2014/041806, каждый из которых подан 10 июня 2014 года; PCT/US 2014/041808, поданную 11 июня 2014 года; и PCT/US 2014/62558, поданную 28 октября 2014 года, и в патенты США №: 61/915148, 61/915150, 61/915153, 61/915203, 61/915251, 61/915301, 61/915267, 61/915260 и 61/915397, поданных 12 декабря 2013 года; 61/757972 и 61/768959, поданные 29 января 2013 года и 25 февраля 2013 года; 61/835936, 61/836127, 61/836101, 61/836080, 61/835973 и 61/835931, поданные 17 июня 2013 года; 62/010888 и 62/010879, поданные 11 июня 2014 года; 62/010329 и 62/010441, поданные 10 июня 2014 года; 61/939228 и 61/939242, каждая из которых была опубликована 12 февраля 2014 года, 61/980012, поданную 15 апреля 2010 года; 62/038358, поданную 17 августа 2014 года; 62/054490, 62/055484, 62/055460 и 62/055487, поданные 25 сентября 2014 года, и 62/069243, поданную 27 октября 2014 года. Ссылка делается на временную заявку США 61/930214, поданную 22 января 2014 года
[829] Упоминается также заявка США 62/180 709, поданная 17 июня 2015 года, PROTECTED GUIDE RNAS (PGRNAS); заявка США 62/091455, поданная 12 декабря 2014 года, PROTECTED GUIDE RNAS (PGRNAS); заявка США 62/096708, поданная 24 декабря 2014 года, PROTECTED GUIDE RNAS (PGRNAS); заявка США 62/091462, поданная 12 декабря 2014 года, DEAD GUIDES FOR CRISPR TRANSCRIPTION FACTORS; заявка США 62/096324, поданная 23 декабря 2014 года, 62/180681, 17 июня 2015 года, и 62/237496, 5 октября 2015 года, DEAD GUIDES FOR CRISPR TRANSCRIPTION FACTORS; заявка США 62/091456, 12 декабря 2014 года и 62/180692, 17 июля 2015 года, ESCORTED AND FUNCTIONALIZED GUIDES FOR CRISPR-CAS SYSTEMS, заявка США 62/091461, 12 декабря 2014 года, DELIVERY, USE AND THERAPEUTIC APPLICATIONS OF THE CRISPR-CAS SYSTEMS AND COMPOSITIONS FOR GENOME EDITING AS TO HEMATOPOETIC STEM CELLS (I ISC's); заявка США 62/094903, 19 декабря 2014 года, UNBIASED IDENTIFICATION OF DOUBLE-STRAND BREAKS AND GENOMIC REARRANGEMENT BY GENOME-WISE INSERT CAPTURE SEQUENCING; заявка США 62/096761, 24 декабря 2014 года, ENGINEERING OF SYSTEMS, METHODS AND OPTIMIZED ENZYME AND GUIDE FUNCTIONAL-CRISPR COMPLEXES; заявка США 62/087475, 4 декабря 2014 года, FUNCTIONAL SCREENING WITH OPTIMIZED FUNCTIONAL CRISPR-CAS SYSTEMS; заявка США 62/055487, 25 сентября 2014 года, FUNCTIONAL SCREENING WITH OPTIMIZED FUNCTIONAL CRISPR-CAS SYSTEMS; заявка США 62/087546, 4 декабря 2014 года, и 62/181687, MULTIFUNCTIONAL CRISPR COMPLEXES AND/OR OPTIMIZED ENZYIFUNCTIONAL-CRISPR COMPLEXES; и заявка США 62/098285, 30 декабря 2014 года, CRISPR MEDIATED IN VIVO MODELING AND GENETIC SCREENING OF TUMOR GROWTH AND METASTASIS.
[830] Упоминаются следующие заявки США 62/181659, 18 июня 2015 года, и 62/207318, 19 августа 2015 года, ENGINEERING AND OPTIMIZATION OF SYSTEMS, METHODS, ENZYME AND GUIDE SCAFFOLDS OF CAS9 ORTHOLOGS AND VARIANTS FOR SEQUENCE MANIPULATION. Упоминаются заявки США 62/181675, 18 июня 2015 года и номер патентного реестра - 46783.01.2128, поданные 22 октября 2015 года, NOVEL CRISPR ENZYMES AND SYSTEMS, заявка США 62/232067, 24 сентября 2015 года, заявка США 62/205733, 16 августа 2015 года, заявка США 62/201542, 5 августа 2015 года, заявка США 62/193507, 16 июля 2015 года, и заявка США 62/181739, 18 июня 2015 года, каждая из которых озаглавлена NOVEL CRISPR ENZYMES AND SYSTEMS, а также в заявке США 62/245270, 22 октября 2015 года, NOVEL CRISPR ENZYMES AND SYSTEMS. Также упоминается заявка США 61/939256, 12 февраля 2014 года, и WO 2015/089473 (PCT/US 2014/070152), 12 декабря 2014 года, каждая из которых озаглавлена ENGINEERING OF SYSTEMS, METHODS AND OPTIMIZED GUIDE COMPOSITIONS WITH NEW ARCHITECTURES FOR SEQUENCE MANIPULATION. Также упоминаются PCT.AJS 2015/045504, 15 августа 2015 года, заявка США 62/180699, 17 июня 2015 года, и заявка США 62/038358, 17 августа 2014, каждая из которых озаглавлена GENOME EDITING USING CAS9 NICKASES.
[831] Каждый из этих патентов, патентных публикаций и заявлений и всех документов, цитируемых в нем или во время их делопроизводства ("приложенные к делу документы"), и всех документов, цитируемых или упоминаемых в приведенных в данном приложении документах, вместе с любыми инструкциями, описаниями, спецификациями и технологическими картами продуктов для любых продуктов, упомянутых в нем или в любом документе и включенных в настоящее описание посредством ссылки, включены в настоящее описание в качестве ссылки и могут быть использованы в практике изобретения. Все документы (например, эти патенты, патентные публикации и заявки и приведенные в приложении документы) включены сюда путем отсылки так же как если бы каждый отдельный документ был специально и индивидуально указан для включения в качестве отсылки.
[832] Кроме того, упоминается заявка PCT PCT/US 14/70057, номер патентного реестра 47627.99.2060 и BI-2013/107, озаглавленная "DELIVERY, USE AND THERAPEUTIC APPLICATIONS OF THE CRISPR-CAS SYSTEMS AND COMPOSITIONS FOR TARGETING DISORDERS AND DISEASES USING PARTICLE DELIVERY COMPONENTS" (притязающая на приоритет по одной или более или всех предварительных заявок на патент США: 62/054490, поданная 24 сентября 2014 года, 62/010441, поданная 10 июня 2014 года, и 61/915118, 61/915215 и 61/915148, каждая из которых подана 12 декабря 2013 года) ("PCT для доставки частиц"), включенной в настоящее описание в качестве отсылки, в отношении способа получения sg-РНК и,tkrf Cas9, включающего смешивание смеси, содержащей sg-РНК и белок Cas9 (и необязательно матрицу HDR) со смесью, содержащей или состоящей только из или состоящей из поверхностно-активного вещества, фосфолипида, биоразлагаемого полимера, липопротеина и спирта; и частицы из такого процесса. Например, когда белок Cas9 и sg-РНК смешивали вместе при подходящем молярном соотношении, например, от 3:1 до 1:3 или от 2:1 до 1:2 или 1:1 при подходящей температуре, например 15-30°С, например, 20-25°C, например, комнатной температуре, в течение подходящего времени, например 15-45, например 30 минут, преимущественно в стерильном, свободном от нуклеазы буфере, например IX PBS. Отдельно компоненты частиц, такие как или содержащие: поверхностно-активное вещество, например, катионный липид, например, 1,2-диолеоил-3-триметиламмонийпропан (DOTAP); фосфолипид, например димиристоилфосфатидилхолин (DMPC); биодеградируемый полимер, такой как полимер этиленгликоля или ПЭГ, или липопротеин, такой как липопротеин низкой плотности, например холестерин, были растворены в спирте, преимущественно C1-6-алкиловом спирте, таком как метанол, этанол, изопропанол, например 100% этанол. Оба раствора смешивали вместе до образования частиц, содержащих комплексы Cas9-sg-РНК. Соответственно, sg-РНК может быть предварительно скомбинирована с белком Cas9 перед формированием всего комплекса в частице. Препараты могут быть получены с другим молярным соотношением различных компонентов, которые, как известно, способствуют доставке нуклеиновых кислот в клетки (например, 1,2-диолеоил-3-триметиламмоний-пропан (DOTAP), 1,2-дитретраканоил-s-глицеро-3-фосфохолин (DMPC), полиэтиленгликоль (ПЭГ) и холестерин). Например, молярные соотношения DOTAP: DMPC: ПЭГ: холестерина могут быть DOTAP 100, DMPC 0, ПЭГ 0, холестерин 0; или DOTAP 90, DMPC 0, ПЭГ 10, холестерин 0; или DOTAP 90, DMPC 0, ПЭГ 5, холестерин 5. DOTAP 100, DMPC 0, ПЭГ 0, холестерин 0. Настоящее описание соответственно охватывает смешение sg-РНК, белка Cas9 и компонентов, которые образуют частицу; а также частиц от такого смешивания. Варианты настоящего изобретения могут включать частицы; например, частицы, используемые в способе, аналогичном способу PCT доставки частиц, например, путем смешивания смеси, содержащей sg-РНК и/или Cas9, как в настоящем изобретении, и компонентов, которые образуют частицу, например, как в для PCT доставки частиц, образуют частицу и частицы из такой смеси (или, конечно, другие частицы, содержащие sg-РНК и/или Cas9, как в настоящем изобретении).
[833] Данное изобретение будет дополнительно проиллюстрировано в следующих примерах, которые приведены только для иллюстративных целей и не предназначены для ограничения изобретения каким-либо образом.
Пример 1: Происхождение и эволюция систем адаптивного иммунитета
[834] Классификация и аннотация систем CRISPR-Cas в геномах архей и бактерий. Локусы CRISPR-Cas имеют более чем 50 семейств генов, при этом не существует строго универсальных генов, происходит быстрая эволюция и наблюдается значительное разнообразие архитектуры локусов. Поэтому не существует возможного древа и комплексный подход особенно необходим. Сейчас существует подробная идентификация cas-генов в рамках 395 профилей для 93 cas белков. Классификация включает профили экспрессии генов и профили архитектуры локусов
[835] Последняя классификация систем CRISPR-Cas представлена на фиг. 1. Класс l включает мультисубъединичные эффекторные комплексы cr-РНК (Cascade), а класс 2 включает односубъединичные эффекторные комплексы cr-РНК (Cas-9 типа). Фиг. 2 представляет молекулярную организацию CRISPR-Cas. Фиг. 3 демонстрирует структуры эффекторных комплексов I и III типов: они имеют общую структуру/ общую природу несмотря на значительные различия в последовательностях. Фиг. 4 показывает CRISPR-Cas как систему, основанную на мотивах узнавания РНК (RRM). На фиг. 5 представлена филогения cas1, где рекомбинация адаптационного и cr-РНК эффекторого модулей показывают основной аспект эволюции систем CRISPR-Cas. На фиг. 6 представлен перечень систем CRISPR-Cas, в особенности распределение типов и подтипов CRISPR-Cas среди архей и бактерий.
[836] Белок Cas1 не всегда связан только с системами CRISPR-Cas, поэтому возможно, что существуют две ветви отдельных "соло" Cas 1, что говорит о том, что возможны различия как в функционировании, так и в происхождении, а также возможно обнаружение новых мобильных элементов (см. Makarova, Krupovic, Koonin, Frontiers Genet 2014). Геномная организация трех семейств казпозонов может предоставить возможные ответы. Вдобавок к cas 1 и PolB, казпозоны имеют различные гены включая нуклеазы (Krupovic et al. BMC Biology 2014). Одно семейство имеет праймированную белком полимеразу, другое семейство имеет РНК-праймированную полимеразу. В добавок к представителям типов Euryarchaeota и Thaumarchaeota, казпозоны, обнаруженные в нескольких бактериях предполагают горизонтальный перенос генов. Казпозон Cas1 (транспозаза/интеграза) предполагает наличие общей базальной филогенетической группы в развитии Cas1.
[837] Бактерии и археи используют CRISPR - систему для адаптивной иммунной защиты в клетках про- и эукариот посредством геномной манипуляции. Белок cas 1 представляет собой уже существующий способ геномной манипуляции. Существуют похожие механизмы интеграции в каспозоны и CRISPR, в особенности репликационно-заваисимое приобретение посредством процессов копирования и вставки, а не вырезания и вставки (Krupovic et al. BMC Biology 2014). Cas1 - есть эталонный образец интегразы (Nunez JK, Lee AS, Engelman A, Doudna JA. Integrase-mediated spacer acquisition during CRISPR-Cas adaptive immunity. Nature. 2015 Feb 18). Есть определенное сходство между терминальными обратными повторами каспозонов и врожденным локусом иммунитета (Koonin, Kmpovic, Nature Rev Genet, 2015). Эволюция систем адаптивного иммунитета в прокариотах и животных возможно шла параллельными путями при помощи интеграции транспозонов в локусы врожденного иммунитета (Koonin, Krupovic, Nature Rev Genet, 2015). Транспозаза RAG1 (ключевой энзим V(D)J-рекомбинации позвоночных) возможно произошел из транспозонов класса Transib (Kapitonov VV, Jurka J.. RAG1 core and V(D)J recombination signal sequences were derived from Transib transposons.. PLoS Biol. 2005 Jun; 3(6):el81), однако ни один из транспозонов класса Transibs не кодирует белок RAG2. RAG1 и RAG2 кодирующие транспозоны описаны в Kapitonov, Koonin, Biol Direct 2015 и филогения транспозазы класса Transib представлена в Kapitonov, Koonin, Biol Direct 2015. Защитное уничтожение ДНК в инфузориях эволюционировало из транспозона PiggyMAc, что вместе с РНК-i образуют врожденный иммунитет (Swart EC, Nowacki M. The eukaryotic way to defend and edit genomes by sRNA-targeted DNA deletion. Ann N Y Acad Sci. 2015).
[838] Относительная стабильность классификации означает, что наиболее часто встречающиеся варианты CRISPR-Cas уже известны. Однако существование редких и до сих пор не включенных в классификацию вариантов также говорит о том, что дополнительные типы и подтипы все ждут своего описания (Makarova et al, 2015. Evolutionary classification of CRISPR-Cas systems and cas genes).
[839] Транспозоны играют ключевую роль в эволюции адаптивного иммунитета и других систем, участвующих в изменении ДНК. Класс 1 CRISPR-Cas возник из транспозонов, однако это касается только адаптационного функционального модуля системы. Класс 2 CRISPR-Cas имеет и адаптационную и эффекторную функции, модули которых могут происходить из разных транспозонов.
Пример 2: Новые предсказанные системы CRISPR-Cas класса 2 и доказательства их независимого происхождения из транспозонов
[840] Системы CRISPR-Cas адаптивного иммунитета бактерий и архей демонстрируют огромное разнообразие состава Cas белков, а также разновидной структурой геномных локусов. Однако эти системы разделяются в целом на два класса: Класс 1 (Class 1), имеющий мульти-протеиновый (многоблоковый) эффекторный комплекс и Класс 2 (Class 2), где эффекторный комплекс состоит из одного большого белка (одноблоковый комплекс), например белка cas-9 (фиг. 1А и 1В). Заявители разработали простую биоинформатическую методику (фиг. 7) для обработки все время пополняемых геномных и метагеномных баз данных, сопровождающихся углублением понимания систем CRISPR-cas для поиска возможных кандидатов для системы CRISPR-Cas класса 2. Анализ базы данных полных (полностью отсеквенированных) геномов при помощи разработанной нами вычислительной методики привело к индентефикации трех новых вариантов, каждый из которых представлен в различных бактериях и имеет в своем составе гены cas1 и cas2, а также третий ген, кодирующий большой белок, который как предполагается функционирует как эффекторный модуль.
В первом из этих локусов, возможный эффекторный белок (C2c1p) имеет похожий на RuvC науклеазный домен и напоминает ранее описанный белок Cpf1, предсказанный эффектор для типа V CRISPR -cas систем, соответственно новая предсказанная система классифицируется как подтип V-B. Всестороннее сравнение белковых последовательностей показало, что эффекторные белки Cas9 Cpf1 и C2Cip, содержащие домен RuvC, возникли из различных групп кодируемых транспозонами TnpB-белков. Вторая группа новых кандидатов для локусов CRISPR-Cas включает большой белок, содержащий два HEPN домена с предсказанной РНК-азной активностью. Учитывая новизну предсказанного эффекторного белка, эти локусы были причислены к новому VI типу CRISPR-Cas, который наиболее вероятно способен к нацеливанию на РНК. В целом, результаты анализа показывают, что системы CRISPR-Cas класса 2 возникли различными, часто не зависимыми друг от друга путями, путем соединения различных кодирующих Cas1-Cas2 адаптационных модулей с эффекторными белками, образованными от различных мобильных генетических элементов. Путь эволюции с наибольшей вероятностью привел к появлению различных вариантов систем класса 2, некоторые из которых еще не открыты.
[840] Системы адаптивного иммунитета CRISPR-Cas присутствуют в геномах 45% бактерий и 90% архей и они демонстрируют значительное разнообразие в составе cas белков, их последовательности, а также архитектуры геномных локусов. Согласно архитектуре эффекторных комплексов cr-РНК, эти системы подразделяются на два класса: Класс 1 имеет мультисубъединичный эффекторный комплекс, а Класс 2 - единственный эффекторный белок (фиг. 1A и 1B). Системы класса 1 встречаются намного чаще и более разнообразны, чем системы класса 2. В настоящей классификации Класс 1 представлен 12-ю отдельными подтипами, кодируемыми многочисленным геномами бактерий и архей, а то время как класс 2 включает 3 подтипа система 2 типа и возможный V тип, который в целом можно обнаружить в 10% отсеквенированных бактериальных геномах (вместе с единственным геномом архей включающим возможную систему данного типа). Системы класса 2 обычно содержат только три или четыре гена в опероне cas, а именно пару генов cas1 ~cas2, которые участвуют в адаптации, но не в интерференции, один многодоменный эффекторный белок, который отвечает за интерференцию, но также способствует процессингу и адаптации пре-cr-РНК. Также часто имеется и четвертый ген с еще неизвестными функциями, которые могут встречаться, по меньшей мере, в некоторых системах II типа. В большинстве случаев CRISPR кассета и ген для определенного типа РНК, известного как tracr-РНК (трансактивирующая небольшая cr-РНК), находятся рядом с оперонами класса 2 (Chylinski, 2014). Tracr-РНК частично гомологична повторам в соответствующей области CRISPR кассеты и имеет важное значение для процессинга пре-cr-РНК, которая катализируется RNAse III, распространенным ферментом бактерий, не связанным с локусами CRISPR-cas (Deltcheva, 2011)(Chylinski, 20I4;Chylinski, 2013).
[842] Эффекторный белок второго типа систем CRISPR-Cas - Cas9 был изучен в мельчайших подробностях в отношении его функций и структуры. В различных бактериях, белки Cas9 включают от ~950 до более чем 1600 аминокислот, а размер варьируются от 950 до 1400 аминокислот. Он содержит два типа нуклеазных доменов, а именно RuvC подобный домен (РНК-аза H консервативный домен) и HNH (MrcA подобный консервативный домен) домен (Makarova, 2011). Кристаллическая структура cas9 показывает "двулопастную" организацию белка с явно выраженным участком узнавания и нуклеазной активности, при этом последний связывается и с RuvC и с HNH доменами (Nishimasu, 2014)(Jinek, 2014). Каждый из нуклеазных доменов Cas9 необходим для разрезания одной из цепочек ДНК-мишени (Jinek, 2012; Sapranauskas, 2011). Недавно было показано что Cas9 участвует во всех трех стадиях CRISPR иммунного ответа, то есть не только приводит к расщеплению ДНК-мишени (стадия интерференции) но также участвует в стадиях адаптации и процессинга пре-cr-РНК (Jinek, 2012). Более конкретно было показано, что определенный домен в участке, обладающем науклеазной активностью узнает и связывается с последовательностью, прилегающей к протоспейсеру (PAM) в вирусной ДНК на стадии адаптации (Nishimasu, 2014)(Jinek, 2014)(Heler, 2015; Wei, 2015). На стадии иммунного ответа CRISPR, Cas9 формирует комплекс с белками Cas l и Cas2, которые непосредственно участвуют в встраивании новых спейсеров во всех системах CRISPR-Cas (Heier, 2015; Wei, 2015).
[843] Белок Cas9, соединенный с tracr-РНК недавно стал ключевым инструментом в создании нового поколения способов редактирования геномов и генной инженерии (Gasiunas, 2013; Mali, 2013; Sampson, 2014; Cong, 2015). Такая ценность Cas9 в генной инженерии основывается на том, что во втором типе CRISPR-Cas в отличие от других систем CRISPR-Cas все операции необходимые для узнавания ДНК-мишени и ее последующего расщепления сосредоточены в рамках одного, но большого мультидоменного белка. Эта особенность систем II типа значительно упрощает разработку эффективных инструментов манипуляции генома. Важно и то, что не все варианты cas9 похожи. На сегодняшний момент большая часть работы по искомому белку была выполнена на cas9 из Streptococcus pyogenes но и другие виды могут предложить существенные преимущества. Что касается рассматриваемого случая последние эксперименты с cas9, который на 300 аминокислот короче чем S. pyogenes позволили упаковать в адено-ассоциированный вирусный вектор, что привело к существенному улучшению возможностей использования системы CRISPR- Cas для редактирования генома in vivo (Ran, 2015).
[844] Системы CRISPR-Cas второго типа в настоящее время подразделяются на три подтипа ((II-A, II-B и II-C) (Makarova, 2011) (Fonfara, 2014; Chylinski, 2013; Chylinski, 2014). Помимо генов cas1, cas2 и cas9, которые характерны для всех локусов II типа, подтип II-A характеризуется дополнительным геном, csn2, который кодирует инактивированную АТФазу (Nam, 2011, Koo, 2012, Lee, 2012), роль которой в приобретении новых спейсеров еще изучена слабо (Barrangou, 2007; Arslan, 2013) (Heler, 2015). В системах подтипа II-B отсутствует csn2, но вместо этого содержится ген cas4, который наоборот более типичен для систем типа I и кодирует 5'-3' экзонуклеазу семейства recB, которая способствует накоплению спейсеров путем генерации рекомбиногенных концов ДНК (Zhang, 2012) (Lemak, 2013, Lemak, 2014). Гены cas1 и cas2 подтипа II-B наиболее тесно связаны с соответствующими белками систем I типа CRISPR-Cas, что подразумевает рекомбинантное происхождение этого подтипа (Chylinski, 2014).
[845] Подтип II-C систем CRISPR-Cas демонстрирует минимальное разнообразие и состоит только из генов casl, cas2 и cas9 (Chylinski, 2013; Koonin, 2013; Chylinski, 2014). Примечательно, однако, что в приобретении спейсера Campylobacter jejuni системами типа II-C требуется участие Cas4, кодируемого бактериофагом (Hooton, 2014). Другой отличительной особенностью подтипа II-C является образование некоторых cr-РНК посредством транскрипции, включая транскрипцию из внутренних альтернативных промоторов, что отличается от процессинга, наблюдаемого во всех других описанных экспериментально системах CRISPR-Cas (Zhang, 2013).
[846] Существование типа V систем CRISPR-Cas было недавно предсказано путем сравнительного анализа бактериальных геномов. Эти предполагаемые новые системы CRISPR-Cas представлены в нескольких бактериальных геномах, в частности, из рода Francisella и одной археи Methanomethylophilus alvus (Vestergaard, 2014). Все предполагаемые локусы типа V включают casl, cas2, отдельный ген, обозначенный как cpf1, и CRISPR кассету (Schunder, 20I3) (Макарова, 2015). Cpf1 представляет собой большой белок (приблизительно 1300 аминокислот), который содержит RuvC-подобный нуклеазный домен, гомологичный соответствующему домену Cas9, а также аналог типичного богатого аргинином кластера Cas9. Однако Cpf1 не обладает нуклеазным доменом HNH, который присутствует во всех белках Cas9, и RuvC-подобный домен является прилегает к последовательности Cpf1, в отличие от Cas9, где он содержит длинные вставки, включая домен HNH (Chylinski, 2014; Makarova, 2015). Эти основные отличия в доменных архитектурах Cas9 и Cpf1 предполагают, что системы содержащие Cpf1, следует классифицировать как новый тип. Состав предполагаемых систем типа V подразумевает, что Cpf1 представляет собой одноблоковый эффекторный комплекс, и, соответственно, эти системы относятся ко второму классу систем CRISPR-Cas. Некоторые из предполагаемых локусов типа V кодируют Cas4 и соответственно напоминают подтипы П-В локусов, тогда как другие не имеют Cas4 и, следовательно, аналогичны подтипу II-C.
[847] Недавно было показано, что самыми близкими гомологами белков Cas9 и Cpf1 являются белки TnpB, которые кодируются транспозонами семейства IS605 и содержат RuvC-подобный нуклеазный домен, а также домен цинкового пальца, который имеет аналог в Cpf1. Кроме того, были идентифицированы гомологи TnpB, которые содержат домен HNH, вставленный в RuvC-подобный домен и показывающий высокое сходство последовательностей с Cas9. Роль TnpB в транспозонах остается неопределенной, поскольку было показано, что этот белок не требуется для транспозиции.
[848] Учитывая гомологию Cas9 и Cpf1 и белков, кодируемых транспозонами, заявители предположили, что системы CRISPR-Cas класса 2 могут возникать многократно в результате рекомбинации между транспозоном и локусом casl-cas2. Соответственно, заявители разработали простую вычислительную стратегию для идентификации геномных локусов, которые могут быть кандидатами для вариантов второго класса. Настоящей заявкой Заявители описывают первое применение этого подхода, в результате которого идентифицируются три группы таких кандидатов, два из которых кажутся принадлежащими к различным подтипам типа V, тогда как третий, по-видимому, подходит к VI типу. Новые варианты систем CRISPR-Cas класса 2 представляют очевидный интерес так как могут быть использованы в качестве инструментов для редактирования и регуляции экспрессии.
[849]Стратегия поиска по базам данных для обнаружения новых кандидатов для CRISPR-Cas локуса класса 2. Заявители использовали простой биоинформатический подход для обнаружения новых претендентов для систем CRISPR-Cas класса 2 (фиг. 7. схема). Поскольку подавляющее большинство локусов CRISPR-Cas включает ген cas1 (Makarova, 2011, Makarova, 2015) а последовательность Cas1 является наиболее консервативной для всех белков Cas (Takeuchi, 2012), Заявители предположили, что cas1 является лучшей из возможных зацепок для определения и поиска возможных новых локусов при помощи PSI-BLAST. После обнаружения всех контигов, кодирующих Casl, путем поиска следующих баз данных WGS (содержащий весь геном) и NT (нуклеотидных) в NCBI, кодирующие белок гены были предсказаны с использованием GenemarkS в районе 20 т.п.н. вышележащих и нижележащих последовательностей от гена casl. Эти предсказанные гены были аннотированы с использованием профилей по базе данных NCBI Conserved Domain Database (CDD) и специфических Cas-белковых профилей (Makarova et al., 2015, Nat Rev Microbiol. 2015, doi: 10.1038/nrmiero3569), а CRISPR кассеты были предсказаны с использованием программы PILER- CR. Эта процедура дала возможность определения обнаруженных локусов CRISPR-Cas в рамках уже известных подтипов. Частичные и/или не поддающиеся классификации возможные локусы CRISPR-Cas, содержащие большие (> 500 ак) белки, были выбраны в качестве возможных кандидатов для новых систем класса 2 основываясь на характерном присутствии таких больших одноблоковых эффекторных белков в системах типов II и V (Cas9 и Cpf1, соответственно). Все 63 возможных локуса, обнаруженные с использованием этих критериев (перечислены в таблице, представленной на фиг. 40A-D), анализировались далее каждый в отдельности с использованием программ PSI-BLAST и HHpred. Последовательности белка, кодированные в локусах-кандидатах, далее использовались в качестве запроса для поиска метагеномных баз данных для дополнительных гомологов, при этом обнаруженные длинные контиги анализировались, как указано выше. В этом вычислительном конвейере было получено в общей сложности 53 новых локуса (некоторые из первоначально идентифицированных 63 локусов-кандидатов были отброшены как ложные, в то время как несколько неполных локусов с отсутствующим cas1 были добавлены) с характерными особенностями CRISPR-Cas, систем класса 2 которые, основываясь на природе предсказанных эффекторных белков, могли бы быть классифицированы на три группы локусов (см. Фиг. 8А и 8В, фиг. 9, 14 и 15 и фиг. 41А-В, 42А-В и 43А-В) Хотя и бактериофаги, заражающие бактерии, которые могли бы иметь недавно открытые системы CRISPR-Cas класса 2, на практике неизвестны, для каждой из этих систем авторы изобретения обнаружили спейсеры, которые соответствуют фагам или предсказанным профагам.
[850] Используя вышеописанную вычислительную стратегию, Заявители обнаружили три новые системы CRISPR-Cas класса 2, а именно C2e1 и C2c3, которые классифицируются как подтипы ранее описанного предполагаемого типа V и C2c2, который Заявители приписывают новому предполагаемому VI типу. Заявители представляют несколько стратегий доказательства того, что эти локусы кодируют функциональные системы CRISPR-Cas. Основываясь на сравнении геномов, авторы изобретения определили фаго-специфические спейсеры для каждой из трех предполагаемых новых систем, а также показали, что наборы спейсеров полностью различаются даже в близкородственных бактериальных геномах, что указывает на активный, функционирующий иммунитет. Многие из этих новых систем встречаются в бактериальных геномах, которые не содержат других локусов CRISPR-Cas, что говорит о том, что системы типа V и типа VI могут функционировать автономно. Более того, даже когда другие системы CRISPR-Cas были идентифицированы в одних и тех же геномах, ассоциированные повторяющиеся структуры были явно отличны от других типов в типах V и VI, что свидетельствует об их независимой функциональности.
[851] Предполагаемая система типа V-B. Первая группа возможных локусов, условно обозначенная как C2c1 (класс 2, кандидат 1), представлена в бактериальных геномах четырех основных таксонов, включая Bacilli, Verrucomicrobia, альфа-протеобактерии и дельта-протеобактерии (фиг. 8A-B "Организация полных локусов систем класса 2" фиг. 41А-В). Все локусы C2c1 кодируют слитый белок Cas1-Cas4, Cas2 и большой белок, обозначаемый Заявителями как C2c1p и, как правило, находятся рядом с CRISPR кассетой (фиг. 9, окрестности C2c1, фиг. 41A-B). В филогенетическом дереве Cas1, соответствующий белковый кластер соединяется с системой типа I-U (фиг. 10A и 10B, фиг. 10C-W, дерево Cas1) и является единственным, в котором обнаружено слияние Cas1-Cas4. Длина белков C2c1p, идентифицированных здесь, варьируется 1100 до 1500 аминокислот, например, может состоять примерно из 1200 аминокислот. HHpred поиск обнаруживает значительное сходство между С-концевой частью белков C2c1p и подмножеством белков TnpB закодированных в транспозонах семейства IS6G5 (фиг. 13А и 13С). Напротив, не было обнаружено никакого существенного сходства между C2c1p и Cas9 или Cpf1, которые аналогичны другим группам белков TnpB (Chylinski, 2014) (Makarova, 2015; Makarova, 2015).
Таким образом, доменная архитектура C2c1p аналогична домену Cpf1 и отличается от архитектуры Cas9 (фиг. 13A), хотя все три белка Cas, по-видимому, эволюционировали из семейства TnpB (фиг. 11 "Организация домена семейств 2-го класса"; фиг. 13А). N-концевая область C2c1p не имеет существенного сходства с другими белками. Прогнозирование вторичной структуры указывает на то, что этот регион принимает преимущественно альфа-спиральную конформацию. Два сегмента сходства с TnpB охватывают три каталитических мотива типа нуклеазы, подобной RuvC, с диагностической сигнатурой D..E..D каталитических аминокислотных остатков (Aravind et al, 2000, Nucleic Acids Res, том 28, 3417-3432) (фиг. 12, "Области гомологии TnpB в белках класса 2"); область, соответствующая мостиковой спирали (также известная как богатый аргинином кластер), который в белке Cas9 участвует в связывании cr-РНК; и небольшую область, которая, по-видимому, является аналогом цинкового пальца TnpB (однако в большинстве белков C2c1 отсутствуют Zn-связывающие цистеиновые остатки, что указывает на то, что такие белки не связывают цинк, более того, C2c1 содержит множественные вставки и делеции в этой области, что указывает на функциональную дивергенцию (фиг. 13А, фиг. 13D-H, фиг. 131). Сохранение каталитических остатков (фиг. 13А) явно указывает на то, что гомологичные домены RuvC всех этих белков являются активными нуклеазами. N-концевые области C2c1 не показывают значительного сходства с любыми известными белками. Предсказание вторичной структуры показывают, что N-концевые области белков C2c1 принимают смешанную α/β-конформацию (фиг. 13D-H, фиг. 1 131). Сходство доменных архитектур C2c1p и Cpf1 предполагает, что локусы C2c1 лучше всего классифицировать как подтип VB, при этом локусы кодирующие Cpf1 - как подтип VA.
[852] Несмотря на то, что гены cas1, связанные с этой системой, очень похожи, повторы CRISPR в соответствующих кассетах являются сильно гетерогенными, хотя все они имеют длину 36-37 п. н. и могут быть классифицированы как неструктурированные (энергия фолдинга AG составляет -0,5-4,5 ккал/моль, тогда как высоко палиндромная CRISPR имеют АГ ниже -7 ккал/моль). Согласно схеме CRISPR (Lange, 2013) несколько повторов V-B подтипа имеют некоторую последовательность или структурное сходство с повторами II типа (фиг. 41А-Т). Однако большинство повторов нельзя было отнести к известным семействам последовательностей или структур они могут быть отнесены к четырем из 6 суперклассов.
[853] Рассматривая возможность того, что предположительные CRISPR-Cas подтипа V-B структурно аналогичны системам II типа, Заявители предприняли попытку идентифицировать tracr-РНК в соответствующих геномных локусах.
[854] Сравнение спейсеров CRISPR кассет типа V-B с небезызбыточной базой данных нуклеотидных последовательностей привело к обнаружению нескольких совпадений с различными бактериальными геномами. В частности, один из спейсеров из Alicyclobacillus acidoterrestris и один из спейсеров от Brevibacillus agri соответствовали неопределенным генам в пределах прогнозируемых профагов, интегрированных в соответствующие бактериальные геномы (фиг. 41А-L).
[855] Предполагаемые системы типа VI. Вторая группа потенциальных локусов CRISPR-Cas, обозначенная как C2c2 (класса 2, кандидат 2), была обнаружена в геномах 5 основных бактериальных таксонов, включая альфа-протеобактерии, бациллы, клостридии, фузобактерии и бактероиды (фиг. 8А-В. "Организация полных локусов систем 2 класса" на фиг. 42А-В). Определенное число локусов C2c2 включают cas1 и cas2 гены, а также большой белок (C2c2p), который не демонстрирует сходства последовательностей с C2c1, Cpf1 или Cas9 и CRISPR кассетой; однако, в отличие от C2c1, C2c2p часто кодируется рядом с CRISPR кассетой, но не casl-cas2 (фиг. 15, окрестности C2c2, фиг. 42A-B). Хотя в вычислительной стратегии авторов изобретения изначально идентифицированные локусы C2c2 охватывали гены cas1 и cas2, последующие поиски показали, что большинство таких локусов может состоять только из гена c2c2 и CRISPR-кассеты. Такие, по-видимому, неполные локусы могут либо кодировать дефектные системы CRISPR-Cas, либо могут функционировать в купе с адаптационным модулем, закодированным в ином месте генома, как это было, к примеру обнаружено для некоторых систем типа III (Majumdar et al., 2015, RNA, vol. 21, 1147-1158). В филогенетическом дереве Cas1 белки Cas1 из локусов C2c2 распределены среди двух клад. Первая клада включает Cas1 от Clostridia и расположена в поддереве II типа вместе с небольшой ветвью типа III-А (фиг. 10А и 10B, фиг. 10C-V, дерево Cas1). Второй клад состоит из белков Cas1 из локусов C2c2 Leptotrichia и расположен внутри смешанной ветви, которая в основном содержит белки Cas1 из систем CRISPR-Cas типа III-A. Поиск по базе данных с использованием HHpred и PSI-BLAST не обнаружил сходства последовательностей между C2c2p и другими белками. Тем не менее, проверка множественных выравниваний белковых последовательностей C2c2p привела к идентификации двух строго консервативных мотивов RxxxxH, которые характерны для доменов HEPN (нуклеотид-связывающий участок высших эукариотических и прокариотических организмов) (Anantharaman et al., 2013, Biol Direct, том 8, 15, Grynberg et al., 2003, Trends in biochemical sciences, vol 28, 224-226) (фиг. 11 и фиг. 13В, фиг. 13J-N). Предсказание вторичной структуры белка указывает на то, что эти мотивы находятся в структурных контекстах, совместимых с доменной структурой HEPN, также как и в целом предсказание вторичной структуры для соответствующих части C2c2p. Домены HEPN представляют собой небольшие (-150 а/к) альфа-спиральные домены с различными последовательностями, но с крайне консервативными каталитическими мотивами, для которых была показана или предсказана РНК-азная активность, а также, что они часто связаны с различными системами защиты (Anantharaman, 2013) (фиг. 13B и 16, Мотив HEPN RxxxxH в семействе C2c2). Последовательности доменов HEPN мало консервативны, за исключением каталитического мотива RxxxxH.
Хотя последовательности двух предполагаемых HEPN-доменов C2c2 мало похожи на другие HEPN-домены, за исключением каталитических мотивов RxxxxH, тип домена в значительной степени подкрепляется предсказанием вторичной структуры, которые указывают на то, что каждый мотив находится в рамках совместимых структурных контекстов (фиг. 13B, фиг. 13J-N). Кроме того, предсказанная вторичная структура всей последовательности для каждого предполагаемого домена также согласуется с укладкой домена HEPN (фиг. 13J-N). Таким образом, представляется вероятным, что C2c2p содержит два активных домена HEPN. Домен HEPN не является новым для систем CRISPR-Cas, поскольку он часто ассоциирован с доменом CARF (CRISPR-Associated Rossmann Fold) в белках Csm6 и Csx1, которые присутствуют во многих системах CRISPR-Cas типа III (Makarova, 2014). Эти белки не относятся ни к адаптационным модулям, ни к эффекторным комплексам, но предполагается, что они способны выполнять некоторые вспомогательные, но еще не охарактеризованные функции в родственных системах CRISPR, а именно, они могут быть компонентами связанного модуля иммунитета, который присутствует в большинстве систем CRISPR-Cas, и работает при запрограммированной гибели клеток (апоптозе), а также выполняет регулятивные функции во время CRISPR ответа (Koonin, 2013; Makarova, 2012; Makarova, 2013). Однако C2c2p отличается от Csmo и Csx1 тем, что этот больший белок является единственным общим белком, кодируемым в локусах C2c2, за исключением Cas1 и Cas2. Таким образом, представляется вероятным, что C2c2p является эффектором в предполагаемых новых системах CRISPR-Cas, а HEPN-домены соответственно являются их каталитическими остатками. Вне предсказанных доменов HEPN последовательность C2c2p не обнаруживала сходства с другими белками и, согласно прогнозам, принимала смешанную альфа/бета вторичную структуру без заметного сходства с любыми известными способами фолдинга белка (фиг. 13J-N).
[856] CRISPR кассеты в локусах C2c2 весьма неоднородны, с длиной от 35 до 39 п.н. и неструктурированны (энергия фолдинга от -0,9 до 4,7 ккал/моль). Согласно картированию CRISPR (Lange, 2013), эти CRISPR не принадлежат ни к одному из установленных структурных классов и относятся к 3 из 6 суперклассов. Только CRISPR от Listeria seeligeri можно было приписать семейству последовательностей номер 24, которое обычно ассоциировано с системами типа II-C (фиг. 42A-L).
[857] Анализ спейсеров локусов C2c2 идентифицировал одну 30 нуклеотидную область, идентичную геномной последовательности Listeria weihenstephanensis, и два неполных совпадения с геномами бактериофагов, в частности, спейсер из Listeria weihenstephanensis совпадал с хвостовым геном бактериофага Listeria (фиг.42A-L).
[858] Учитывая уникальность предсказанного эффекторного комплекса C2c2, эти системы могут быть классифицированы как предположительный тип VI систем CRISPR-Cas. Более того, поскольку все экспериментально охарактеризованные и ферментативно активные HEPN-домены являются РНК-азами, системы типа VI вероятно, действуют на уровне РНК, таких как мРНК,
[859] Предполагаемые системы типа V-С. Третья группа локусов-кандидатов включает исключительно метагеномные последовательности и, следовательно, не может быть отнесена к конкретным таксонам. Эти локусы включают только два кодирующих белок гена, которые кодируют Cas1 и большой белок, обозначенный C2c3 (класса 3, кандидат 3) (фиг. 8A "Организация полных локусов систем 2 класса"; фиг. 14, окрестности C2c3, фиг. 44A-Б). Белки C2c3 находятся в том же диапазоне размеров, что и Cpf1 и C2c1, и аналогично содержат TnpB-гомологичный домен на своих C-концах, который, в отличие от соответствующего домена C2c1, продемонстрировал ограниченное, но значительное сходство с Cpf1 (фиг. 13А и 13С). Области гомологичности TnpB C2c3 содержат три каталитических мотива типа RuvC-подобной нуклеазы, с диагностической триадой D..E..D каталитических аминокислотных остатков (Aravind et al., 2000, выше), область, соответствующую мостиковой спирали (также известную как богатый аргинином кластер), который участвует в cr-РНК-связывании в Cas9, и небольшую область, которая представляется соответствующей "цинковому пальцу" TnpB (Zn-связывающие остатки цистеина сохраняются в C2c3). Сохранение каталитических остатков явно свидетельствует о том, что гомологичные домены RuvC всех этих белков являются активными нуклеазами. N-концевые области C2c1 и C2c3 не демонстрируют существенного сходства друг с другом или с любыми известными белками. Предсказание вторичной структуры показывает, что N-концевые области белков C2c3 принимают смешанную конформацию cx/j3. Таким образом, общие архитектуры доменов C2c1 и C2c3 и, в частности, организация домена RuvC аналогичны областям Cpf1, но отличаются от архитектуры Cas9. Это говорит о том, что локусы C2c1 и C2c3 правильнее классифицировать как подтипы V-B (см. выше) и V-C, соответственно, с локусами кодирующими Cpf1 на настоящий момент обозначенными подтипом V-A,
[860] Среди c2c3 локусов только один содержит CRISPR кассету с нехарактерно короткими спейсерами длиной 17-18 нуклеотидов. Повторы в кассетах имеют длину 25 п.н. и оказывается, что они неструктурированные и имеют энергию 1,6 ккал/моль (фиг. 43A-F).
[861] Спейсеры из единственного континга C2c3, содержащего CRISPR кассету, слишком коротки, чтобы иметь статистически значимые совпадения. Тем не менее, было обнаружено несколько совпадений с последовательностями из предсказанных профагов (ФИГ. 43 A-F).
[862] Подтипы белков TnpB имеющие значительное сходство с известным (Cas9) и тремя описанными здесь предполагаемыми эффекторами класса 2 (Cpf1, C2c1 и C2c3) не совпадали (фиг. 13А и 13С). Хотя расхождение в последовательности TnpB-подобных доменов слишком велико для обеспечения надежного филогенетического анализа, эти данные свидетельствуют о том, что четыре известных в настоящее время больших эффекторных белка класса 2, Cas9, Cpf1, C2c1 и C2c3 развивались независимо от генов различных мобильных генетических элементов.
[863] Хотя большинство спейсеров в новых локусах CRISPR-Cas, описанных в настоящем описании, не были в значительной степени похожи на любые доступные последовательности, существование спейсеров, совпадающих с геномом фага, подразумевает, что эти локусы могут кодировать активные функциональные адаптивные системы иммунитета. Малая доля фаго-специфических спейсеров типична для систем CRISPR-Cas и, скорее всего, указывает на их динамическую эволюцию и малую долю вирусного разнообразия, которая представлена в современных базах данных последовательностей. Эта интерпретация соотносится с наблюдением, что близкородственные бактериальные штаммы, кодирующие гомологичные локусы CRISPR-Cas, обычно содержат несвязанные коллекции спейсеров, примером которых являются локусы C2c2 из Listeria weihemtephanensis и Listeria newyorkensis (фиг. 45А-С).
[864] Заявители применили простую и понятную вычислительную стратегию для прогнозирования новых CRISPR-cas систем класса 2. Ранее описанные системы класса 2, а именно тип II и предполагаемый тип V, состояли из генов cas1 и cas2 (а в некоторых случаях и cas4), содержащих адаптационный модуль и один большой белок, который составляет эффекторный модуль. Поэтому заявители предположили, что любой геномный локус, содержащий cas1 и большой белок, может быть потенциальным кандидатом на новую систему класса 2 и заслуживает тщательного исследования. Такой анализ с использованием чувствительных способов сравнения белковых последовательностей привел к идентификации трех наиболее вероятных кандидатов, два из которых являются отдельными подтипами ранее описанного предполагаемого типа V (подтипы VB и VC), тогда как третий может быть классифицирован как новый предполагаемый тип VI, в силу наличия нового предсказанного эффекторного белка. Многие из этих новых систем встречаются в бактериальных геномах, которые не содержат других локусов CRISPR-Cas (фиг. 44А-E), что указывает на возможность автономного функционирования систем типа V и типа VI. Рассматриваемые в настоящем описании локусы-кандидаты были проверены с помощью функциональных способов, которые выявили экспрессию и процессинг соответствующих CRISPR-кассет, производящих зрелые cr-РНК, определили предполагаемую tracr-РНК (где возможно), продемонстрировали интерференцию при экспрессии в E.coli, определили PAM и выявили минимальные компонентов, необходимые для расщепления лизата.
[865] Системы типа V кодируют предсказанные эффекторные белки, которые по общей доменной архитектуре напоминают Cas9, но, в отличие от Cas9, RuvC-подобные домены Cpf1, C2c1 и C2c3 являются соседними и не имеют вставок, характерных для Cas9, в частности домена нуклеазы HNH. Наличие одного вместо двух доменов нуклеазы указывает на то, что эффекторные белки типа V механически отличаются от Cas9, в которых домены HNH и RuvC отвечают за расщепление комплементарных и не комплементарных цепей ДНК-мишени соответственно (Chen et al., 2014, The Journal of biological chemistry7, vol. 289, 13284- 13294, Gasiunas et al., 2012, Proceedings of the National Academy of Sciences of the United States of America, vol. 109, E2579-2586; Jinek et al., 2012, Science, vol. 337, 816-821). Предсказанные белки-эффекторы типа V могут образовывать димеры, в которых два домена, подобные RuvC, могут расщеплять противоположные цепи молекулы-мишени.
[866] В основе предполагаемых систем CRISPR-Cas типа VI по-видимому лежит новый эффекторный белок, который содержит два предсказанных домена HEPN, которые, подобно ранее характеризованным доменам HEPN, могут обладать РНК-азной активностью, что системы типа VI могут быть нацелены на и расщеплять мРНК. Ранее сообщалось о нацеливании на мРНК для некоторых систем типа CRISPR-Cas типа III (Hale et al., 2014, Genes Dev, том 28, 2432-2443, Hale et al, 2009, Cell, vol. 139, 945-956, Peng et al., 2015, Nucleic acid research, vol. 43, 406-417). Альтернативная возможность заключается в том, что C2c2 является первой ДНК-азой в суперсемействе HEPN, возможно, с двумя доменами HEPN, каждый из которых расщепляет одну цепь ДНК. Таким образом, кажется возможным преобразование C2c1 и C2c2 в инструменты редактирования генома с различными классами целей.
[867] Чтобы проверить функциональность этих систем CRISPR-Cas класса 2, заявители показали, что две C2c1 CRISPR-кассеты экспрессируются, процессируются в зрелые cr-РНК и способны к интерференции в случае экспрессии в E. coli. Эти эксперименты выявили более характеристик локуса C2c'l, включая: (i) процессируемые прямые повторы (DR) на 5'-конце cr-РНК, (ii) 5' PAM и (iii) наличие короткой РНК с гомологией повтор-антиповтор по отношению к процессируемому 5' DR, то есть предполагаемая tracr-РНК. Открытие процессируемых DR на 5'-конце cr-РНК и 5' PAM поддерживает такое развитие событий при котором C2c1 происходит из систем класса 1, потому что эти системы демонстрируют процессинг 5'-концов (I типа и III типа) и 5'-концевой последовательности PAM (I типа) (Mojica et al., 2009, Microbiology, vol. 155, 733-740; Makarova et al., 2011, Nat Rev Microbioln, т. 9, 467-477). В данном случае для С2с1 идентифицирована AT-богатая последовательность PAM, в противоположность ГЦ-богатым последовательностям PAM хорошо охарактеризованных систем II класса. Для локусов C2c1, охарактеризованных в настоящем описании экспериментально, Заявители определили cr-РНК, которые обрабатываются до длины, при которой сохраняется возможность к связыванию и кофолдингу с предполагаемыми tracr-PHKs, что указывает на то, что tracr-PHKs могут быть вовлечены и, возможно, даже необходимы для образования комплексов. Затем авторы изобретения использовали экспрессию C2c1 в культуре клеток человека для экспериментальной верификации того, что в этих условиях участвовала tracr-РНК и была необходима для in vitro расщепления ДНК-мишени конкретной исследуемой нуклеазой C2c1.
[868] Заявители также показали, что, когда C2c2 локус из L.seeligeri экспрессируется в E.coli, он процессируется в cr-РНК с прямым повтором (DR) на 5'-конце 29 нуклеотидов длиной, похожие результаты были получены для локуса C2c2 из L.shahii. В этом случае, вырожденный повтор находится в начале последовательности, а не в конце, как это типично для большинства CRISPR-последовательностей, и последовательность и гены cas транскрибируются сонаправленно. Заявители не обнаружили предполагаемую tracr-РНК в данных секвенирования РНК C2c2 РНК. Однако прогнозируемая вторичная структура прямого повтора (DR) 29 нуклеотидов длиной показывает стабильную "шпильку", которая может быть потенциально важной для образования комплекса с эффекторным белком C2c2.
[869] На фиг. 94 показано, что процессинг последовательности C2c2 в E. coli требует белка С2с2, как оценено с помощью in vitro транскрибированных последовательностей спейсеров, инкубированных с белком С2с2.
[870] В сочетании с результатами предыдущих анализов, (Chylinski, 2014; Makarova, 2011), идентификация новых систем CRISPR-Cas класса 2 обнаруживает главную тенденцию в развитии систем CRISPR-Cas класса 2. Эффекторные белки двух или трех типов в этом классе, по-видимому, эволюционировали из пула мобильные генетических элементов, которые кодируют белки TnpB, содержащие RuvC-подобный домен. Последовательности RuvC-подобных доменов TnpB и гомологичных доменов эффекторных белков класса 2 слишком сильно дивергировали для надежного филогенетического анализа. Тем не менее, для Cas9, эффекторного белка системы II Типа, специфичная группа предков, по-видимому, легко идентифицируется, а именно семейство TnpB-подобных белков, особенно обильных в цианобактериях, которые показывают относительно высокое сходство с Cas9 и разделяют с Cas9 всю доменную архитектуру, а именно RuvC-подобные домены, нуклеазные домены HNH и богатую аргинином мостиковую спираль (Chylinski, 2014) (Рисунок 11, Фиг. 13A и 13B, "Доменная организация семейств 2 класса"; Рисунок 12, Фиг. 13A и 13B, "области гомологии TnpB в белках 2 класса"). В отличии от Cas9, невозможно отнести Cpf1, C2c1, и C2c3 к определенному семейству, несмотря на сохранение всех мотивов, сконцентрированных в каталитических остатках RuvC-подобных нуклеаз, эти белки показывают только ограниченное сходство с родовыми профилями TnpB. Однако, учитывая, что C2clp не показывает обнаружимого сходства последовательностей с Cpf1, что Cpf1, C2c1, и C2c3 содержат различные вставки между мотивами RuvC и явно неродственными N-концевыми областями, наиболее вероятно, что Cpf1, C2c1, и C2c3 возникли независимо из различных семейств в пуле кодирующих элементов TnpB.
[871] Любопытно, что белки TnpB, во-видимому, "предопределены" для использования в эффекторных комплексах систем CRISPR-Cas класса 2, так что они, по-видимому, были рекрутированы во множестве разных случаев. По-видимому, такая полезность белков TnpB связана с их предсказанной способностью разрезать одноцепочечную ДНК, будучи связанными с молекулой РНК через R-богатую мостиковую спираль, которая, как это было показано, в Cas9 связывает cr-РНК (Jinek, 2014; Nishimasu, 2014; Anders et al., 2014, Nature, vol. 513, 569-573). Функции TnpB в жизненных циклах соответствующих транспозонов плохо изучены. Эти белки не нужны для транспозиции, и в одном случае было показано, что белок TnpB снижает транспозицию (Pasternak, 2013), но их механизм действия остается неизвестным. Экспериментальное исследование TnpB, вероятно, прольет свет на механические аспекты систем CRISPR-Cas класса 2. Стоит отметить, что механизмы Cpf1 и C2c1 могут быть похожими друг на друга, но они существенно отличаются от Cas9, так как в первых двух белках отсутствует домен HNH, который в Cas9 отвечает за разрыв одной из цепей ДНК-мишени (Gasiunas, 2012)(Jinek, 2012)(Chen, 2014). Соответственно, использование Cpf1 и C2c1 может привести к дополнительным возможностям редактирования генома.
[872] С точки зрения эволюции поразительно, что системы CRISPR-Cas класса 2, по-видимому, полностью произошли из различных мобильных генетических элементов, учитывая недавние данные о вероятном возникновении генов cas1 из отдельного семейства транспозонов (Koonin, 2015, Krupovic, 2014). Более того, вероятное независимое происхождение эффекторных белков из разных семейств TnpB, наряду с различной филогенетической близостью соответствующих cas1 белков, настоятельно свидетельствует о том, что системы класса 2 развивались неоднократно посредством сочетания различных адаптационных модулей и полученных из транспозонов нуклеаз, что привело к появлению эффекторных белков. Этот способ эволюции, по-видимому, является окончательным проявлением модульности, характерной для эволюции системы CRISPR-Cas (Makarova, 2015), что подразумевает, что в природе могут существовать дополнительные комбинации адаптационного и эффекторного модулей.
[873] Предсказанные системы CRISPR-Cas типа VI содержат предсказанный эффекторный белок, в составе которого присутствуют два предсказанных домена HEPN, которые, вероятно, обладают РНК-азной активностью. Эти домены HEPN не являются частями эффекторных комплексов в других системах CRISPR-Cas, но задействованы в осуществления различных защитных функций, включая прогнозируемую вспомогательную роль (Anantharaman, 2013) (Makarova, 2015) в различных системах CRISPR Cas. Присутствие HEPN-доменов в качестве каталитических единиц предсказанного эффекторного модуля подразумевает, что системы типа VI нацеливаются на и расщепляют мРНК. Ранее для систем CRISPR-Cas III Типа уже сообщалось о таргетировании мРНК (Hale, 2014; Hale, 2009) (Peng, 2015). Несмотря на то, что домены HEPN до сих пор не были обнаружены в настоящих транспозонах, они характеризуются высокой горизонтальной подвижностью и являются неотъемлемой частью мобильных элементов, таких как системы токсин-антитоксин (Anantharaman, 2013). Таким образом, предполагаемые системы типа VI, похоже, соответствуют общей парадигме модульной эволюции CRISPR-Cas класса 2 от мобильных элементов, и ожидается, что новые варианты и новые типы систем будут обнаружены путем анализа данных геномики и метагеномики. Учитывая, что белок C2c2 не связан с другими эффекторами класса 2 (все они содержат RuvC-подобные домены, даже при условии, отдаленного родства), открытие типа VI можно считать подтверждением независимого происхождения других вариантов класса 2.
[874] Учитывая вырисовывающийся сценарий эволюции систем класса 2 из мобильных элементов, представляется полезным изучить общую эволюцию локусов CRISPR-Cas и, в частности, вклад мобильных элементов в этот процесс (фиг. 53). Предшественники адаптивной иммунной системы, скорее всего, возникли из-за вставки каспозона (транспозона, кодирующего Cas), примыкающего к локусу, который кодировал примитивную систему врожденного иммунитета (iKoonin и Krupovic, 2015, Nature reviews Genetics, vol. 16, 184-192; Krupovic et al., 2014, BMC Biology, vol. 12, 36). Важным результатом было также включение системы токсин-антитоксин, которая передавала ген cas2 и могла появиться либо в предковом каспазоне, либо в изменяющемся локусе адаптивного иммунитета (фиг. 51).
[875] Учитывая чрезвычайно широкое распространение систем класса 1 в археях и бактериях и распространение в них древних доменов RRM (мотив распознавания РНК, RNA Recognition Motif - RRM), не возникает сомнений, что предковая система принадлежала к классу 1 (фиг. 51). Скорее всего, предковая архитектура напоминала существующий тип III и включала ферментативно активный белок Cas10 (Makarova et al, 2011, Biol Direct, том 6, 38; Makarova et al, 2013, Biochem Soc Trans, том 41, 1392-1400). Белок Cas10 представляет собой гомолог ДНК-полимераз семейства В и нуклеотидных циклаз семейства GGDEF, который показывает значительное сходство последовательностей с этими ферментами и сохраняет все каталитические аминокислотные остатки (Makarova et al., 2011, Biol Direct, том 6, 38, Makarova et al., 2006, Biol Direct, том 1, 7). Структурный анализ подтвердил наличие в Cas10 полимеразоциклазоподобного домена и дополнительно выявил второй, вырожденный и, по-видимому, неактивный домен этого семейства (Khachatryan et al., 2015, Phys Rev Lett, vol. 114, 051801; Shao et al., 2013, Structure, vol. 21, 376-384, Zhu and Ye, 2012, FEBS Lett, том 586, 939-945). Точный характер каталитической активности Cas10 остается неясным, но было показано, что каталитические остатки полимеразоциклазоподобного домена являются существенными для расщепления ДНК-мишени (Saniai et al., 2015, Cell, vol. 161, 1164-1174). Белки Cas8, присутствующие в системах типа CRISPR-Cas I Типа, сходны по размерам с Cas10 и занимают эквивалентные положения в эффекторных комплексах (Jackson et al., 2014, Science, vol. 345, 1473-1479; Jackson and Wiedenheft, 2015, Mol Cell, vol. 58, 722-728, Staals et al., 2014, Molecular cell, vol. 56, 518-530), что наводящий на размышления об эволюционном родстве между большими субъединицами эффекторных комплексов III Типа и I Типа. Более конкретно, белки Cas8, которые сильно расходятся в последовательности между подтипами I Типа, могут быть каталитически неактивными производными Cas10 (Makarova et al., 2011, Biol Direct, том 6, 38, Makarova et al., 2015). Этот сценарий предполагает правдоподобную направленность эволюции, от подобного типу III предкового системы класса 1 до систем типа I. Дивергенция систем типа III и типа I могла быть ускорена за счет получения гелиазы Cas3 во время возникновения типа I (фиг. 53). Различные типы и подтипы класса 2 затем эволюционировали посредством множественных замещений блока гена, кодирующего эффекторные комплексы класса 1, путем введения транспозируемых элементов, кодирующих различные нуклеазы (фиг. 53). Эта конкретная направленность эволюции вытекает из наблюдения того, что адаптационные модули разных вариантов класса 2 происходят из разных типов 1-го класса (фиг. 10А и 10В).
[876] Системы класса 2 CRISPR-Cas, по-видимому, полностью получены из разных мобильных элементов. В частности, по-видимому, было, по меньшей мере, два (в подтипе V-C), но обычно три или, в случае II Типа, даже четыре представителя мобильных элементов: (i) предковый каспозон, (ii) модуль токсин-антитоксин, который дал начало Cas 2 (iii) транспозируемый элемент, во многих случаях кодирующий TnpB, который был предком эффекторного комплекса класса 2, и (iv) в случае II Типа нуклеаза HNH могла быть пожертвована предковому транспозону с помощью самосплайсирующегося интрона группы I или группы II (Stoddard, 2005, Q Rev Biophys, vol. 38, 49-95) (фиг.53). Локусы предполагаемого типа V-C, описанные в настоящем описании, кодируют конечную минимальную систему CRISPR-Cas, единственную в настоящее время идентифицированную, которой не хватает Cas2; по-видимому, сильно расходящиеся подтипы V-C Cas1-белков способны самостоятельно формировать адаптационный комплекс без вспомогательной субъединицы Cas2. Многократное возникновение систем класса 2 из мобильных элементов представляет собой окончательное проявление модульности, характерное для эволюции CRISPR-Cas (Makarova et al., 2015).
[877] Демонстрация того, что различные разновидности систем класса 2 CRISPR-Cas, независимо от того, что они эволюционировали из разных транспозонов, подразумевает, что дополнительные варианты и новые типы еще предстоит идентифицировать. Хотя большинство, если не все новые системы CRISPR-Cas, как ожидается, будут редкими, они могут использовать новые стратегии и молекулярные механизмы и могут стать важным ресурсом для новых, разнообразных приложений в области инженерии генома и биотехнологии.
[878] Модульная эволюция является ключевой особенностью систем CRISPR-Cas. Этот способ эволюции, по-видимому, наиболее выражен в системах класса 2, которые развиваются благодаря сочетанию адаптационных модулей от различных других систем CRISPR-Cas с эффекторными белками, которые, как представляется, рекрутируются из мобильных элементов в нескольких независимых случаях. Учитывая экстремальное разнообразие мобильных элементов в бактериях, представляется вероятным, что эффекторные модули систем класса 2 CRISPR-Cas также очень разнообразны. Здесь заявители использовали простой вычислительный метод для определения трех новых вариантов систем CRISPR-Cas, но многие другие, вероятно, будут содержаться в бактериальных геномах, которые еще не были секвенированы. Хотя большинство, если не все из этих новых систем CRISPR-Cas, как ожидается, будут редкими, они могут использовать новые стратегии и молекулярные механизмы и станут важным ресурсом для новых приложений в области инженерии генома и биотехнологии.
[879] Программа TBLASTN со значением отсечки 0,01 и отключенной фильтрацией низкой сложности была использована для поиска для профиля Cas1 (Makarova et al., 2015) в качестве запроса к базе данных NCSI WGS. Последовательности контигов или полных геномов, в которых был обнаружено попадание (результат с высокими баллами), были извлечены из той же базы данных. Область вокруг гена Cas1 (область 20 т.п.н. от начала гена Cas1 и 20 т.п.н. от конца гена Casl) была извлечена и транслирована с использованием GeneMarkS (Besemer et al, 2001, выше). Для предсказанных белков каждой Casl-кодирующей области производился поиск с использованием набора профилей из базы данных CDD (Marchler-Bauer, 2009) и конкретным профилям белков Cas (Makarova et al., 2015) с использованием программы RPS-BLAST (Marchler-Bauer et al, 2002, Nucleic Acids Res, том 30, 281-283). Процедура определения полноты локусов CRISPR и классификации систем CRISPR-Cas в существующие типы и подтипы (Makarova et al., 2015), разработанные ранее, были применены к каждому локусу.
[880] CRISPRmap (Lange, 2013) использовался для повторной классификации.
[881] Частичные и/или неклассифицированные локусы, которые охватывали белки размером более 500 аминокислот, анализировали на индивидуальной основе. В частности, для каждого предсказанного белка, кодируемый этими локусами, был произведен поиск с использованием итеративных поисков профилей с PSI-BLAST (Altschul, 1997), и композиционная статистика с отключенной фильтрацией низкой сложности для поиска удаленных аналогичных последовательностей против базы данных "безызбыточных" (NR) белков NCBF. Для каждого идентифицированного "безызбыточного" белка производился поиск в базе данных WGS с использованием программы TBLAST (Altschul, 1997). Программа HHpred использовалась с параметрами по умолчанию для идентификации сходства отдаленной последовательности (Soding, 2005), используя в качестве запросов все белки, идентифицированные в поисках BLAST. Множественные выравнивания последовательностей были построены с использованием MUSCLE (Edgar, 2004) и MAFFT (Katoh and Standiey, 2013, Mol Biol Evol, vol, 30, 772-780). Филогенетический анализ проводился с использованием программы FastTree с эволюционной моделью WAG и дискретной гамма-моделью с 20 категориями скоростей (Price et al., 2010, PLoS One, том 5, e9490). Вторичная структура белка была предсказана с использованием Jpred 4 (Drozdetskiy, 2015).
[882] Повторы CRISPR были идентифицированы с использованием PILER-CR (Edgar, 2007, supra) или, для вырожденных повторов, CRISPRfinder (Grissa et at., 2007, Nucleic Acids Res, vol.35, W52-57). Программа Mfold (Zuker, 2003, Nucleic Acids Res, том 31, 3406-3415) была использована для определения наиболее стабильной структуры повторяющихся последовательностей. Для спейсерных последовательностей производился поиск по базам данных нуклеотидов NCBI NR и WGS с использованием MEGABLAST (Morgulis et at., 2008, Bioinformatics, том 24, 1757-1764) с использованием параметров по умолчанию, за исключением размера слова был установлен равным 20.
[882] CRISPR-повторы были обнаружены при помощи PILER-CR (Edgar, 2007, выше) или для вырожденных повторов - CRISPRfinder (Grissa et al., 2007, Nucleic Acids Res, vol. 35, W52-57). Программа Mfold (Zukler, 2003, Nucleic Acids Res, vol 31, 3406-3415) были использованы для обнаружения наиболее стабильных структур для повторяющихся последовательностей.
[883] Был выполнен поиск последовательностей спейсеров был выполнен по базам данных NCBI: NR and WGS database при помощи MEGABLAST (Morgulis et al., 2008, Bioinformatics, vol. 24, 1757-1764) с заданными параметрами по умолчанию за исключением того, что параметр wordsize был задан на 20
[884] Выбранные кандидаты генов
[885] ГeнID: A;
Тип гена:C2C1;
Организм: 5. бактерия Opitutaceae TAV5;
Длина спейсера - мода (диапазон): 34 (33-37);
DR1: 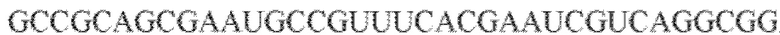 ;
;
DR2: нет;
tracr-РНК1:
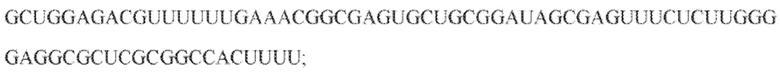
tracr-РНК2: нет;
Последовательность белка:


[886] ID гена: B;
Тип гена: C2C1;
Организм: 7. Bacillus thermoamylovorans штамма B4166;
Длинаспейсера - мода (диапазон): 37 (35-38);
DR1: 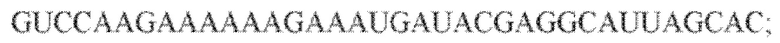
DR2: нет;
tracr-РНК1; 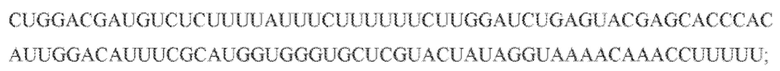
tracr-РНК2: нет;
Последовательность белка:

[887] ID гена: C;
Тип гена: C2CI;
Организм: 9. Bacillus sp. NSP2.1;
Длина спейсера - мода (диапазон): 36 (35-42);
DR1: 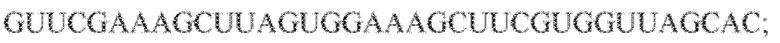
DR2: нет;
tracr-РНК1:
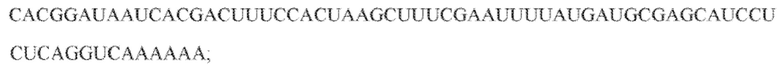
tracr-РНК2: нет;
Последовательность белка:

[888] ID гена: D;
Тип гена: C2C2;
Организм: 4. Бактерия Lachnospiraceae NK4A144 G619;
Длина спейсера - мода (диапазон): 35;
DR1: 
DR2: 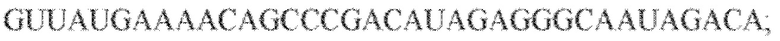
tracr-РНК1: нет;
tracr-РНК2: нет;
Последовательность белка:

[889] ID гена: E;
Тип гена: C2C2;
Организм: 8. Listeria seeligeri серовар 1/2b штамм SLCC3954;
Длина спейсера - мода (диапазон): 30;
DR1: 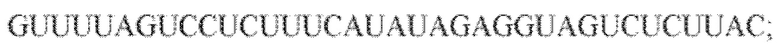
DR2: нет;
tracr-РНК1:
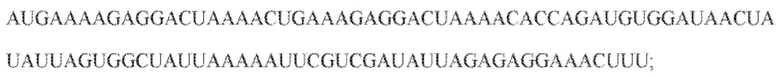
tracr-РНК2: нет;
Последовательность белка:

[890]
ID гена: F;
Тип гена: C2C2;
Организм: 12. Leptotrichiawadei F0279;
Длина спейсера - мода(диапазон): 31;
DR1: 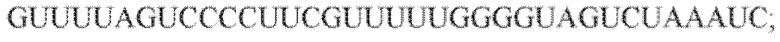
DR2: нет;
tracr-РНК1:
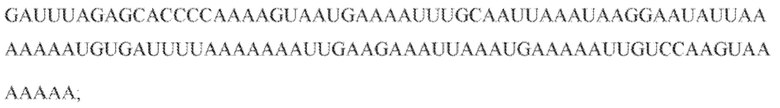
tracr-РНК2:
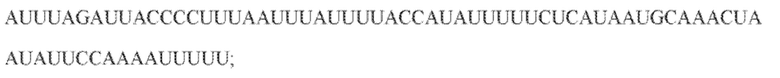
Последовательность белка:

[891] ID гена: G;
Тип гена: C2C2;
Организм: l4. Leptotrichiashahii DSM 19757 B031;
Длина спейсера - мода (диапазон): 30 (30-32);
DR1: 
DR2: нет;
tracr-РНК1: 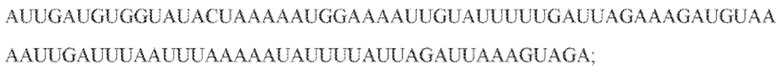
tracr-РНК2: нет;
Последовательность белка:


[892] ID гена: H;
Тип гена: Cpf1;
Организм: Francisellaularensis subsp. novicida U112,
Длина спейсера - мода (диапазон): 31;
DR1: 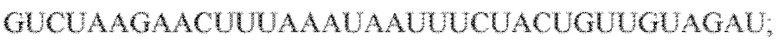
DR2: нет;
tracr-РНК1:
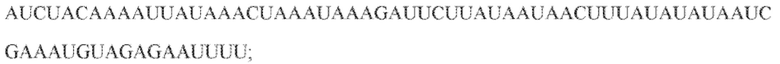
tracr-РНК2: нет;
Последовательность белка:


[893] Гены для синтеза
[894] Для генов от А-Н заявители оптимизируют гены для экспрессии в человеке и добавляют следующую последовательность ДНК в конец каждого гена. Обратите внимание, что эта последовательность ДНК содержит стоп-кодон (подчеркнутый), поэтому стоп-кодон не добавляется к последовательности, оптимизированной кодоном гена:
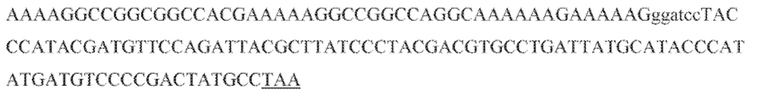
[895] Для оптимизации избегайте следующих сайтов рестрикции: BamHI, EcoRI, HindIII, BsmBI, BsaI, BbsI, AgeI, XhoI, Ndel, NotI, Kpnl, BSrGI, SpeI, XbaI, NheI
[896] Эти гены клонированы в простой экспрессирующий вектор млекопитающих:
[897] A
[898] 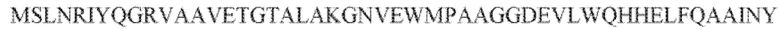


[899] B
[900] 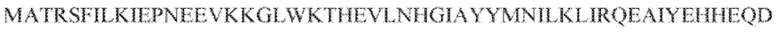

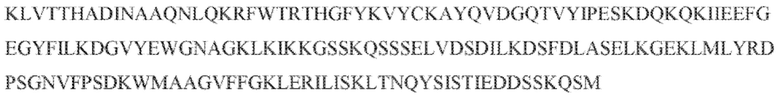
[901] C
[902] 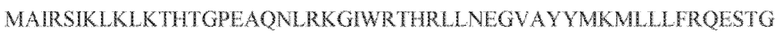

[903] D
[904] 


[905] E
[906] 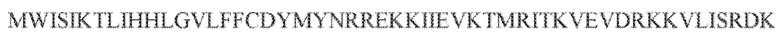


[907] F
[908] 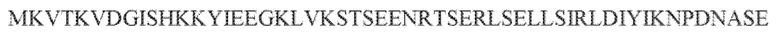

[909] G
[910] 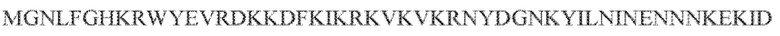
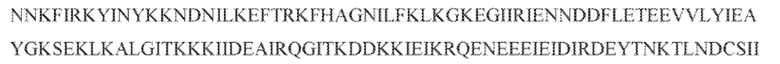

[911] H
[912] 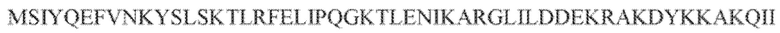


[913] Локусы от A до G клонированы и вставлены в плазмиду с низким количеством копий. Используется вектор, не содержащий имеет резистентности к Amp.
[914] A-локус
[915] 





[916] В-локус
[917] 




[918] C-локус
[919]





[920] D-локус
[921]







[922] Е-локус
[923]




[924] F-локус
[925]




[926] G-локус
[927]




Пример 3: Дальнейшая оценка C2c2p и ассоциированных компонентов
[928] Заявители выделили оба локуса С2с2 из Carnobacterium gallinarum (фиг. 46) и изучили доменную структуру и организацию, а также экспрессию последовательности CRISPR. В первом локусе С2с2 наблюдается низкая экспрессия двух последовательностей CRISPR в направлении гена С2с2 (См. Фиг. 47). Второй локус также имеет низкую экспрессию транскрипции в направлении гена С2с2 (См. Фиг. 48). Заявители определили, что оба локуса могут иметь минимальную экспрессию, поскольку ни один локус не имеет ассоциированных генов Cas (Cas1/Cas2). Такие связанные гены могут быть необходимы для функционального локуса CRISPR. Заявители оптимизировали способы получения локусов C2c2 из других бактериальных штаммов.
[929] Заявители выполняют секвенирование РНК следующих культивируемых штаммов: Clostridium aminophilum DSMI0710, Carnobacterium gallinarum DSM4847, Leptotrichia wade F0279, Leptotrichia shahii DSM19757 и Rhodobacter capsulatus SB1003.
[930] Заявители разрабатывают pACYC клонирование из следующих источников ДНК. Претенденты клонируют весь локус С2с2 в E.coli - основу для экспериментов по разрезанию ДНК/РНК.
1) Выделенная геномная бактериальная ДНК: Lachnospiraceae bacterium MA2020, Lachnospiraceae bacterium NK4A179, Lachnospiraceae bacterium NK4A144
2) ДНК из культивируемого штамма: Clostridium aminophilum DSM10710, Carnobacterium gallinarum DSM4847, Leptotrichia wade F0279, Leptotrichia shahii DSM19757, Rhodobacter capsulatus SB1003
[931] Претенденты разрабатывают библиотеки фрагментов PAM для разрезания ДНК для оценки способности к разрезанию эффекторного белка С2с2. Претенденты разрабатывают эксперименты по разрезанию РНК на основе разрезания транскрипта гена устойчивости.
[932] Заявители тестируют функцию в клетках млекопитающих с использованием продуктов PCR промотора U6: спейсер (DR-спейсер-DR) (в некоторых аспектах спейсеры могут относиться к cr-РНК или направляющей РНК или аналогичным термином, как написано в этой заявке) и tracr для следующих штаммов: Lachnospiraceae bacterium, Listeria seeligeri serovar 12b, Leptotrichia wadei, Leptotrichia shahii. Кодирующую белок C2c2 ДНК Заявители оптимизировали кодоном для экспрессии в млекопитающих, и клонировали в плазмиду из Genscript.
[933] Заявители проанализировали репрезентативный локус С2с2, т.е. CRISPR локус C2c2 Listeria seeligeri серовара 1 /2b штамма SLCC3954 (LseC2c2). Заявители выполняли секвенирование РНК локуса C2c2 L. seeligeri, который был клонирован в E. coli. Локус LseC2c2 был синтезирован плазмидами Genscript в pET-28 вектор. Клетки, содержащие плазмиды, были сделаны компетентными при помощи Z-компонентного набора (Zymo). E. coli, содержащая гетерологичные конструкции, была культивирована в среде Luria broth с добавлением соответствующих антибиотиков в суспензию при 37°C и 300 об/мин. Бактерии выращивали в аэробных условиях и собирали в стационарной фазе роста.
[934] РНК была выделена из бактерий в стационарной фазе, сначала бактерии были ресуспендированы в TRIzol, а затем гомогенизированы с шариками циркония/кремния (BioSpec Products) в BeadBeater (BioSpec Products) в течение 7 одноминутных циклов. Общая РНК была очищена из гомогенизированных образцов с помощью протокола Miniprep Direct-Zol RNA (Zymo), обработаны ДНК-азой TURBO DNAase (Life Technologies), и 3'-дефосфорилирована T4-полинуклеотид-киназой (New England Biolabs). рРНК удаляли с помощью набора для удаления бактериальной рРНК Ribo-Zero (Illumina). Библиотеки секвенирования РНК были получены из рРНК-обедненной РНК с использованием модификации ранее описанного способа секвенирования cr-РНК (Heidrich et al., 2015, Methods Mol Biol, vol. 1311, 1-21). Кратко, транскрипты получили поли-А-хвост с помощью поли-(A)-полимеразы E. coli (New England Biolabs), были лигированы с 5'-РНК-адаптерами с использованием T4 РНК-лигазы 1 (оцРНК-лигаза), высокой концентрации (High Concentration, New England Biolabs) и обратно транскрибированы многотемпературной обратной транскриптазой AffmityScript (Agilent Technologies). кДНК была амплифицирована ПЦР с помощью штрих-кодированных праймеров с использованием полимеразы Herculase II (Agilent Technologies).
[935] Готовые библиотеки кДНК секвенировали на MiSeq (Alumina). Считывания из каждого образца были идентифицированы на основе ассоциированных с ними штрих-кодов и выровнены в соответствии с эталонным геномом RefSeq с использованием BWA (Li and Durbin, 2009, Bioinformatics, vol. 25, 1754-1760). Попарные выравнивания были использованы для выделения последовательностей целых транскриптов с помощью инструментов Picard (http://broadinstitute.github.io/picard) и эти последовательности были проанализированы с использованием Geneious 8.1.5.
[936] Заявители наблюдали высокий уровень экспрессии в локусе и образование мелких cr-РНК с прямыми повторами (DR) на 5'-конце длиной 29 нуклеотидов и спейсерами длиной 15-18 нуклеотидов (фиг. 49A). Хотя локус LseC2c2 содержит предсказанную предполагаемую tracr-РНК (фиг. 15), Заявители не наблюдали его экспрессии (фиг. 49A). Эти данные свидетельствуют о том, что вторичная структура, присутствующая в pre-cr-РНК локуса LseC2c2 может быть достаточной для процессинга для получения зрелой cr-РНК, а также загрузки cr-РНК на белок C2c2. РНК-фолдинг процессированной cr-РНК демонстрирует надежно предсказанную шпильку внутри прямого повтора, которая потенциально может служить в качестве "ручки" для белка C2c2 (фиг. 49А).
[937] Заявители также экспрессировали локус C2c2 в Leptotrichia shahii str. SLCC3954 в E. coli и анализировали его экспрессию с использованием Нозерн-блоттинга. Процедура проводилась, по существу, как описано в Pougach and Severinov, 2012 (Methods Mol Biol, vol. 905, 73-86). Клетки E. coli BL21 AI трансформировали плазмидой pACYCduet-1, содержащей контролируемый T7-промотором cas operon Leptotrichia shahii, и плазмидой pCDF-lb, содержащей минимальную кассету CRISPR с одним спейсером. Общая РНК была экстрагирована из 5 мл клеток E.coli, индуцированных 1 мМ арабинозы/0,2 мМ IPTG и выращенных до OD600 0,8-1,0. Клетки лизировали с помощью 5-минутной обработки с использованием Max Bacterial Enhancement Reagent с последующей очисткой РНК реагентом TRIzol (Thermo Fisher Scientific). 15 пг общей РНК разделяли в денатурирующей 8 М мочевине - 12% полиакриламидном геле и электрофоретически переносили на мембрану Hybond-XL (GE Healthcare) с использованием Mini Trans-Blot Electrophoretic Transfer Cell (Bio-Rad). Мембрану высушивали, а затем подвергали кросс-линкингу ультрафиолетовым излучением. Раствор для гибридизации ExpHyb (Clontech) использовали для гибридизации в соответствии с инструкциями производителя в течение 1 часа при 40°C с олигонуклеотидными зондами с 32P-мечеными концами. Заявители обнаружили, что матрица CRISPR экспрессируется и процессируется в cr-РНК длиной 44 п.н. (фиг. 49B), экспрессию и образование cr-РНК как показано здесь, по меньшей мере, в двух различных локусах C2c2, используя независимые способы.
[938] Заявители стремились предсказать потенциальные tracr-РНК для остальной части идентифицированных локусов C2c2 путем поиска последовательностей анти-повторов в каждом локусе. Во многих локусах CRISPR-Cas повтор, расположенный на дальнем от промотора конце последовательности CRISPR, является вырожденным и имеет последовательность, которая явно отличается от остальных повторов (Biswas et al., 2014, Bioinformatics, vol. 30, 1805 -1813). Такие вырожденные повторы были обнаружены в нескольких системах C2c2 и C2c1, что позволило Заявителям предсказать направление транскрипции последовательности. Интегрируя эту информацию, были идентифицированы предполагаемые tracr-РНК для 4 из 17 локусов C2c2 и 4 из 13 локусов C2c1. В некоторых подтипах II-В и II-С последовательности CRISPR транскрибируется в противоположном направлении, начиная с вырожденного повтора (Sampson et al., 2013, Nature, vol. 497, 254-257; Zhang et al, 2013, Mol Cell, vol. 50, 488-503). Соответственно, авторы изобретения попытались предсказать tracr-РНК в разных положениях по отношению к последовательности CRISPR, но не смогли идентифицировать дополнительные последовательности tracr-РНК-кандидатов. Разумеется, предсказание tracr-РНК для других локусов было затруднено из-за сочетания таких факторов, как неполная комплементарность повторов, отсутствие связанной последовательности CRISPR и/или потенциальная неполнота локусов. Кроме того, существует вероятность того, что не все системы CRISPR класса 2 требуют tracr-РНК.
[939] Заявители идентифицировали обедненные мотивы последовательности, чтобы идентифицировать нуклеотиды последовательностей PAM. Библиотека PAM была перенесена в бактериальный вектор для трансформации штамма E.coli, экспрессирующего LshC2c2 (фиг. 54). Более подробный анализ для РНК-мишени приведен ниже, при условии, что для прямого распознавания требуется последовательность PAM. В этом анализе используют два штамма E.coli. Один из них несет плазмиду, кодирующую эндогенный локус эффекторного белка из бактериального штамма. Другой штамм несет пустую плазмиду (например, PACYC184, контрольный штамм). Все возможные последовательности PAM 7 или 8 п.н. представлены на плазмиде резистентности к антибиотику (pUC19 с геном устойчивости к ампициллину). Последовательность PAM расположена рядом с последовательностью прото-спейсера 1 (мишень РНК к первому спейсеру в эндогенном локусе эффекторного белка). Были клонированы две библиотеки PAM. Одна из них имеет 8 случайных п.н. на 5'-конце протоспейсера (например, всего 65536 различных последовательностей PAM=сложность). Другая библиотека имеет 7 случайных п.н. на 3'-конце протоспейсера (например, общая сложность составляет 16384 различных последовательностей PAM). Обе библиотеки были клонированы так, чтобы иметь в среднем 500 плазмид на каждую возможную последовательность PAM. Тестовый штамм и контрольный штамм трансформировали библиотеками 5'-PAM и 3'-PAM в отдельных трансформациях, а трансформированные клетки высевали отдельно на планшетах с ампициллином. Распознавание и последующее разрезание/интерференция в плазмиде делает клетки чувствительными к ампициллину и предотвращает рост. Примерно через 12 ч после трансформации все колонии, образованные тестовыми и контрольными штаммами, были отобраны и выделена плазмидная РНК. Плазмидную РНК использовали в качестве матрицы для амплификации ПЦР и последующего глубокого секвенирования. Представленность всех последовательностей PAM в нетрансформированных библиотеках показала ожидаемую представленность PAM в трансформированных клетках. Представленность всех PAM, обнаруженных в контрольных штаммах, показало фактическую представленность. Представленность всех PAM в тестируемом штамме показала, что PAM не распознаются ферментом, а сравнение с контрольным штаммом позволяет получить обедненную последовательность PAM. Интерференция CRISPR приводит к неэффективной трансформации плазмидами, содержащими эффективную последовательность-мишень. Трансформированные плазмиды секвенировали для идентификации нецелевых последовательностей. Обедненные последовательности идентифицируют 5'-PAM-нуклеотиды (фиг. 55). Гетерологичное нацеливание таргетинг в E. coli наблюдалось для трех мишеней. Повышенная интерференция наблюдалась для более активно транскрибированных мишеней. В частности, мишень в транскрибируемой области ("РНК") совпала с увеличением интерференции по сравнению с минимально транскрибируемой мишенью "ДНК1" и "ДНК2" (фиг. 56). На 5'-PAM-скрининге ДНК не было обнаружено расщепления ДНК (фиг. 85). LshC2c2 не расщепляет нетранскрибируемую или транскрибируемую ДНК в анализе in vitro с использованием E. coli RNAP (фиг. 83). LshC2c2 не расщепляет оцДНК или дцДНК in vitro (фиг. 84).
[940] Компоненты LshC2c2 очищали для испытаний in vitro (фиг. 57). Первоначально в тестовых реакциях наблюдалось, что cr-РНК расщепляется C2c2. Расщепление cr-РНК не зависит от Mg2+, и может быть увеличено при отсутствие мишени (фиг. 58). Кроме того, было обнаружено, что в отсутствие Mg (фиг. 108) наблюдается уменьшение расщепления.
[941] Показывая способность локуса LshC2c2 CRISPR опосредовать интерференцию оцРНК, авторы изобретения хотели продемонстрировать два дополнительных аспекта активности C2c2: 1) Интерференция РНК с использованием ортогонального анализа и 2) способность перенацелить (ретаргетировать) C2c2 на эндогенно экспрессируемые транскрипты в клетке. Авторы изобретения разработали флуоресцентный способ считывания активности LshC2c2 путем экспрессии RFP из трансфицированной плазмиды в E. coli (фиг. 63A). Затем авторы изобретения разработали три спейсера для каждой из трех возможных H-PAM (всего 9 спейсеров), нацеленных на мРНК RFP, и клонировали их в остов pLshC2c2, как и раньше. Авторы изобретения трансфицировали этими плазмидами E. coli, уже экспрессирующую плазмиду RFP и выращивали в течение ночи в условиях двойной селекции. Анализируя уровни RFP в E.coli с помощью проточной цитометрии, авторы изобретения наблюдали надежный нокдаун RFP для всех трех PAM и отсутствие нокдауна RFP для спейсеров, нацеливающих на антисмысловую цепь ДНК, или ненацеливающих спейсеров (фиг. 63B-C). Для дальнейшего исследования активности LshC2c2 в отношении нацеливания и расщепления, спейсеры, нацеливающие на RFP, клонировали в локус LshC2c2, и локус экспрессировали в E. coli, несущей плазмиду, кодирующую экспрессируемый RFP, или контрольную плазмиду pUC19. На фиг. 61 показано, что C2c2 нацелен на транскрибированный RFP. Зависимость интерференции от цепи была исследована путем выбора последовательностей-мишеней, совпадающих с транскрибированными областями или комплементарными им. Высокие уровни интерференции наблюдались с использованием нацеливающих последовательностей, комплементарных транскрибированной РНК (фиг. 62). Также наблюдалось, что степень интерференции варьирует между транскрибируемыми мишенями, возможно, связано с уровнями транскрипции (фиг. 63).
[942] Нацеливание на РНК и выбор цели были изучены с использованием модельной транскрипции и тестирования различных целей (фиг. 65). Расщепление РНК было протестировано с помощью РНК-мишеней (фиг. 66 и 67) и РНК, транскрибируемой с матрицы ДНК (фиг. 68 и 69). Наблюдаемые продукты расщепления РНК совпадали по размеру с ожидаемыми от модельного транскрипта (фиг. 70). Фиг. 87 демонстрирует, что LshC2c 2 не требует малой РНК для расщепления РНК.
Пример 4: C2C2 нацеливается на и разрезает РНК in vitro
C2c2 Leptrichia shahii способен к интерференции в оцРНК фага MS2
[943] C2c2 был впервые обнаружен при компьютерном поиске консервативных неизвестных белков вблизи адаптационного белка Cas2, чтобы выявить новые системы CRISPR класса 2 (Shmakov et al) и предположительно является функциональным эффектором нового подтипа VI CRISPR из-за небольшой гомологии с другими известными белками CRISPR. Белки C2c2 имеют два консервативных домена HEPN, которые демонстрируют сильную консервативность активных остатков, но мало гомологичны с другими известными белками суперсемейства домена HEPN или эффекторами CRISPR. Однако C2c2 отличается от других белков HEPN, в частности CRISPR-ассоциированных белков типа Csx1 и Csm6, которые обычно димеризуются до расщепления РНК, поскольку у них есть два домена HEPN, а не один. Многие из этих уникальных особенностей привели к классификации C2c2 как предполагаемого типа VI. Учитывая эти наблюдения и распространенность белков C2c2-семейства у разных видов бактерий, авторы изобретения попытались определить, являются ли C2c2 биологически активными локусами CRISPR-Cas и могут ли опосредовать интерференцию против РНК.
[944] Чтобы определить, является ли C2c2 (LshC2c2) Leptotrichia shahii функциональной нацеленной на РНК системой, авторы изобретения клонировали весь локус LshC2c2 CRISPR-Cas в низкокопийные плазмиды (pLshC2c2), чтобы обеспечить гетерологичную репарацию в E. coli. В охарактеризованных на настоящий момент нацеленных на ДНК и РНК системах CRISPR расщепление мишеней зависит от двух факторов: 1) комплементарности между спейсерной последовательностью cr-РНК и целевым сайтом (протоспейсером) и 2) наличием соответствующего прилегающего мотива (PAM), фланкирующего протоспейсер. В связи с необходимостью для PAM отчетливого разделения "своего" и "чужого", неясно, необходима ли системе специфичного нацеливания на РНК последовательность PAM, поскольку тогда, по-видимому, не будет ауто-РНК для нацеливания.
[945] Чтобы исследовать требования к PAM и активность LshC2c2, авторы изобретения использовали анализ рестрикции фага MS2 (фиг. 73). Фаг MS2 является идеальной моделью для исследования расщепления РНК, поскольку он представляет собой литический одноцепочечный РНК-содержащий фаг, который не имеет ДНК-интермедиатов в жизненном цикле. Он легко заражает E. coli путем прикрепления к F-пилям и, таким образом, может использоваться для тестирования гетерологичной интерференции в сравнении с оцРНК. Авторы изобретения синтезировали библиотеку последовательностей cr-РНК для разбивки каждого возможного целевого участка длиной 28 п.н. в геноме фага MS2, чтобы определить, какие целевые участки были значительнее обеднены, чем другие. Библиотека cr-РНК была клонирована в pLshC2c2, так что каждый уникальный спейсер был первым спейсером в последовательности из двух спейсеров. Авторы изобретения трансформировали этой библиотекой E. coli NovaBlue (DE3, F +) и выращивали культуры с фагом MS2 или без него в течение ночи. Используя этот анализ, авторы изобретения смогли идентифицировать однонуклеотидную PAM, анализируя фланкирующие области целевых последовательностей cr-РНК, которые были обогащены из-за устойчивости к инфекции MS2. Анализ выявил 3'H-PAM (не G-PAM) на РНК-мишени, что указывает на то, что комплекс LshC2c2 избирателен в отношении последовательности (фиг. 75А-В). Помимо идентификации PAM, скрининг показал, что гетерологично экспресированный локус LshC2c2 способен к значительной интерференции с оцРНК и защите от инфекции фагом MS2.
[946] Чтобы проверить результаты скрининга, авторы изобретения клонировали четыре наиболее обогащенных спейсера и показали от 3- до 4-log-кратное снижение эффективности образования бляшек в соответствии с уровнем обогащения, наблюдаемым в скрининге. Более того, авторы изобретения хотели еще раз подтвердить обнаружение PAM, и поэтому клонировали серию из четырех направляющих молекул на один возможный нуклеотид PAM (всего 16 направляющих молекул), все нацеливающие на область гена mat MS2. Авторы изобретения обнаружили, что было осуществлено эффективное нацеливание на все 16 мишеней были с более сильным предпочтением C, A и U. Поскольку G-PAM, тем не менее, были нацеленными, и их меньшинство было обогащено в скрининге интерференции, этот PAM может быть более релаксированным, чем 3'-H PAM.
[947] Белок C2C2 из Leptotrichia shahii был экспрессирован в E. coli и очищен с использованием гистидиновой метки (His-tag) с тремя последующими раундами гель-фильтрации на Akta FPLC с использованием колонки Superdex 200. Для экспериментов по расщеплению in vitro 175-нуклеотидная РНК-мишень (меченые t1 и t3 соответственно, последовательности см. ниже) объединяли с 5-кратным молярным избытком белка C2c2 и cr-РНК (с использованием 28-нуклеотидных спейсеров и 28-нуклеотидных прямых повторов, последовательности см. ниже) и инкубировали при 37°С в течение 15 минут в буферах, указанных на рисунке рядами. Реакцию останавливали инкубированием протеиназой К в течение 15 мин при 37°С и затем денатурировали в загрузочном буфере для TBE-мочевины при 85°С в течение 5 мин. Образцы анализировали на денатурирующем геле TBE Urea PAGE.
[948] Результаты показывают, что C2C2 опосредует эффективную деградацию РНК-мишени зависимым от cr-РНК образом. (фиг. 71, фиг. 79, фиг. 80). Кроме того, сама cr-РНК также расщепляется во время этого процесса.
[949] Нацеливание на транскрипты RFT у бактерий показало, что скорость роста снижается (фиг. 77). Без связи с теорией, это может указывать на то, что система HEPN представляет собой суицидальную систему защиты фагов.
[950] Фрагменты расщепления были отображены, как показано на фиг. 81 и 114.
[951] Секвенирование РНК IVC (расщепление in vitro) проводили, как показано на фиг.82.
[952] Последовательность РНК-мишени 1
[953] 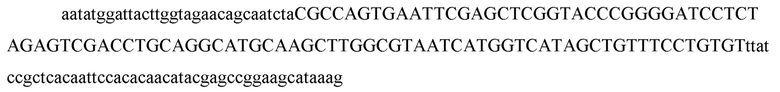
[954] Последовательность РНК-мишени 3
[955] 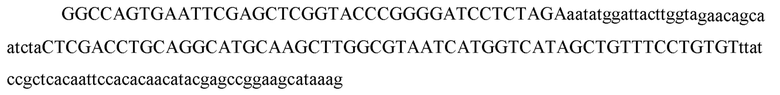
[956] Последовательность cr-РНК
[957] CCACCCCAATATCGAAGGGGACTAAAACtagattgctgttctaccaagtaatccat
[958] Последовательность белка C2c2 L.shahii
[959] 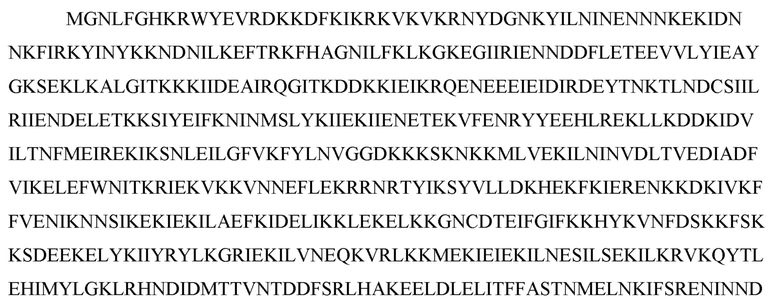

[960] Скрининг РНК PAM-последовательностей, идентифицированных с использованием интерференции фага MS2, см. Фиг. 73-78. Было определено, что LshC2c2 имеет 3'-PAM для расщепления РНК (фиг. 86). Фиг. 88 и 89 также демонстрируют, что LshC2c2 является перепрограммируемым и чувствительным к PAM.
[961] Фиг. 90 демонстрирует, что LshC2c2 не может использовать спейсеры меньше 18-22 нуклеотидов.
[962] Фиг. 91-93 показывают влияние шпильки (ее модификаций) на активность по расщеплению. Показано, что без шпильки cr-РНК не расщепляются (фиг. 91) и что в целом если в стебле и возможны замены отдельных оснований, то активность нарушается только при изменениях вторичной структуры. Эксперименты по укорочению DR также указывают на то, что разрушение стеблевой структуры предотвращает расщепление (фиг. 92 и фиг. 93).
[963] Фиг. 100 показывает влияние двухвалентных катионов на активность C2c2.
[964] Было показано, что C2c2 отщепляет 3'-конец от целевого сайта (фиг. 102 и фиг. 103)
[965] На фиг. 104-106 показано, что C2c2 можно перепрограммировать с помощью cr-РНК.
[966] Не ограничиваясь теорией, по-видимому, будучи активированным, C2c2 становится активным и деградирует другие РНК (фиг. 98, фиг. 108).
[967] Было показано, что C2c2 может быть перепрограммирован с помощью cr-РНК (фиг. 104-106) и что нацеливание может осуществляться на длинные мишени (фиг. 107)
[968]
[969] Фиг. 112 предполагает, что cr-РНК C2c2 имеют seed-последовательность, о чем свидетельствует анализ одиночного и двойного нарушения комплементарности. Действительно, двойное нарушение комплементарности в нуклеотидах 1-11 мишени заметно влияет на расщепление, однако в меньшей степени, если находится в области, охватывающей нуклеотиды 16-26. Эти цифры также указывают на специфичность расщепления эффекторного белка C2c2.
[970] Фиг. 113 и фиг. 114, предполагают, что изменения контекстов последовательности влияли на паттерн расщепления. В действительности целевые последовательности, предоставленные в другом контексте, расщепляются по-другому.
Пример 5: Мутация в любом из доменов HEPN устраняет направленное расщепление
[971] Были созданы варианты Cpc2, содержащие мутации R597A и R1278A. Как показано на фиг. 72, обе мутации прекращают расщепление РНК, см. Также фиг. 97, который демонстрирует, что R597A, 11602Л, R1278A и H1283A прекращают расщепление РНК
[972] Мутанты по домену HEPN продолжают процессировать естественную последовательность (фиг. 95).
Пример 6:
[973] Мутанты по домену HEPN сохраняют целевую связывающую активность, как показано на фиг. 111 (анализ EMSA). Верхний ряд: связывание дикого типа C2c2. Нижний ряд: связывание R1278A с мутантным Lsh C2c2.
[974] Соответствующие остатки в других ортологах C2c2 были идентифицированы структурным выравниванием для идентификации структурных представителей, которые соответствуют либо их экспериментально определенным структурам, либо гомологическим моделям. Фиг. 109 иллюстрирует выравнивание последовательностей следующих ортологов Leptotrichia shahii DSM 19757 C2c2; Rhodohacter capsulatus SB 1003 (RcS); Rhodobacter capsulatus R121 (RcR); Rhodohacter capsulatus 1) 17442 (RcD); Lachnospiraceae bacterium MA2020 (Lb (X)); Lachnospiraceae bacterium NK4A179 (Lb (X), [Clostridium] aminophilum DSM 10710 (CaC), Lachnospiraceae bacteriumNK4A144 (Lb (X), Leptotrichia wadei F0279 (Lew), Leptotrichia wadei F0279 (Lew), Carnobacterium gallinarum DSM 4847 (Cg); Carnobacterium gallinarum DSM 4847 (Cg), Paludibacter propionicigenes WB4 (Pp), Listeria seeligeri serovar l/2b (Ls), Listeria weihenstephanensis FSL R9-0317 (Liw) и Listeria bacterium FSL M6-0635 (Lib). Фиг. 110 демонстрирует, что ортологи C2c2 сохраняют домены HEPN.
[975] Использование нумерации из консенсусной последовательности, полученной с использованием выравнивания MUSCLE (www.ebi.ac.uk/Tools/msa/muscle/) были идентифицированы следующие консервативные остатки: K36, КЗ9, V40, E479, L514, V518, N524, G534, K535, E580, L597, V602, D630, F676, L709, I713, R717 (HEPN), N718, H722 (HEPN), E773, P823, V828, I879, Y880, F884, Y997, L1001, F1009, L1013, Y1093, L1099, L1111, Y1114, L1203, D1222, Y1244, L1250, L1253, K1261, I1334, L1355, L1359, R1362, Y1366, E1371, R1372, D1373, R1509 (HEPN), H1514 (HEPN), Y1543, D1544, K1546, K1548, V1551, 11558. Парное совпадение консервативных остатков в консенсусной последовательности с аминокислотами Leptotrichia wadei C2c2 (последовательность F здесь) представляет собой: K36,K2; K39,K5; V40,V6; E479,E301; L514,L331; V518J335; N524,N341; G534,G351; K535,K352; E580,E375; L597,L392; V602,L396; D630,D403; F676,F446; L709,I466; 1713,1470; R717 (HEPN),R474; N718,H475; H722 (HEPN),H479; E773,E508; P823,P556; V828,L561; 1879,1595; Y880,Y596; F884,F600; Y997,Y669; L1001,I673; F1009,F681; L1013,L685; Y1093,Y761; L1099,L676; L1111,L779; Y1114,Y782; L1203,L836; D1222,D847; Y1244,Y863; L1250,L869; L1253,I872; K1261,K879; 11334,1933; L1355,L954; L1359,I958; R1362,R961; Y1366,Y965; E1371,E970; R1372,R971; D1373,D972; R1509 (HEPN),R1046; H1514 (HEPN), H1051; Y1543,Y1075; D1544,D1076; K1546,K1078; K1548,K1080; V1551,I1083; 11558,11090.
Пример 7: Создание мутантов C2c2 с повышенной специфичностью
[976] Недавно был описан способ создания ортологов Cas9 с повышенной специфичностью (Slaymaker et al., 2015). Эта стратегия может быть использована для повышения специфичности ортологов C2c2. Первичные остатки для мутагенеза - все положительно заряженные остатки в пределах домена HEPN, так как это единственная известная структура в отсутствие кристалла, и известно, что специфичные мутанты по RuvC работали в Cas9. Консервативными остатками аргинина в домене HEPN являются R717 и R1509.
[977] Дополнительные кандидаты представляют собой положительно заряженные остатки, консервативные среди различных ортологов, таких как K2, K39, K535, K1261, R1362, R1372, K1546 и K1548. Они могут быть использованы для получения мутантов C2c2 с повышенной специфичностью.
Пример 8: C2c2 представляет собой однокомпонентный программируемый направленный на РНК РНК-нацеливающий эффектор CRISPR
Таблица А. Последовательности cr-РНК, использованные для экспериментов in vitro.
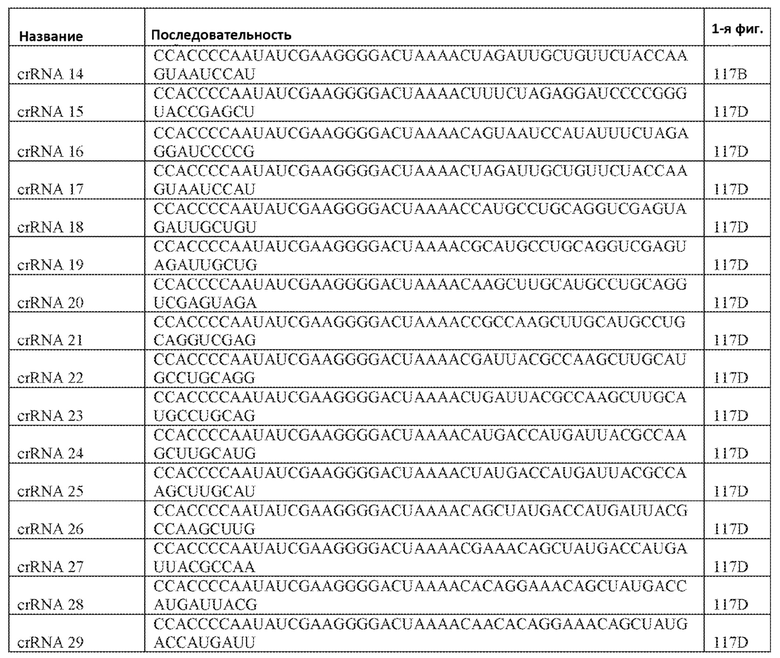
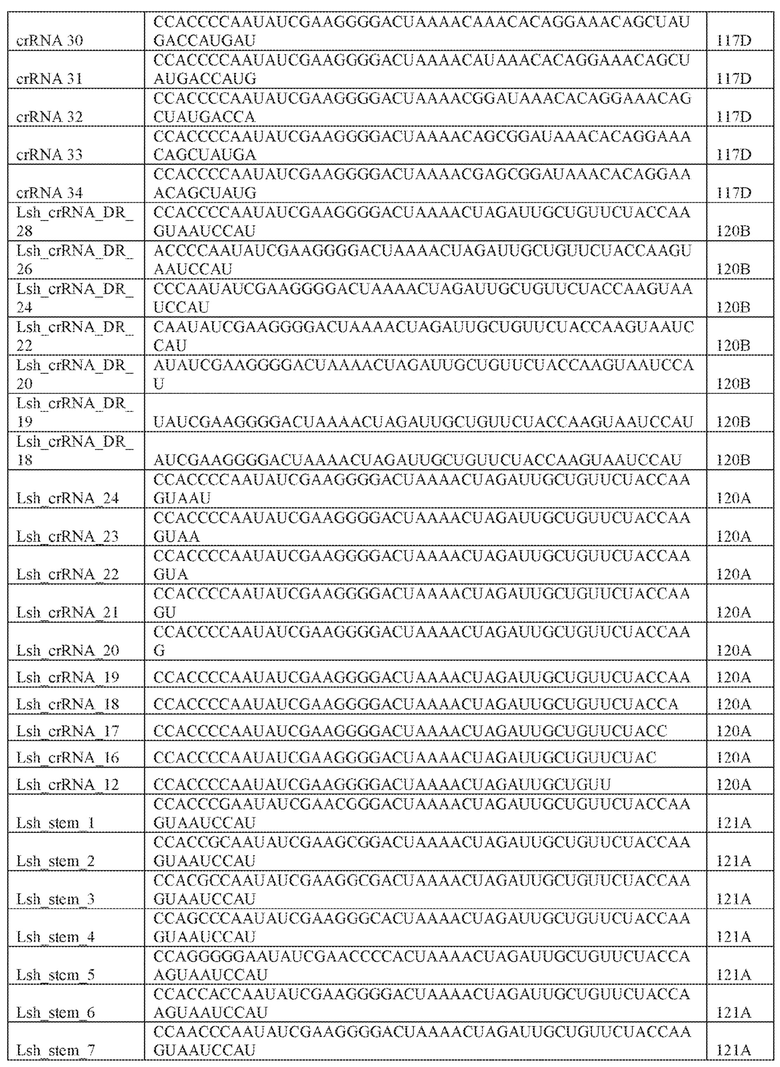
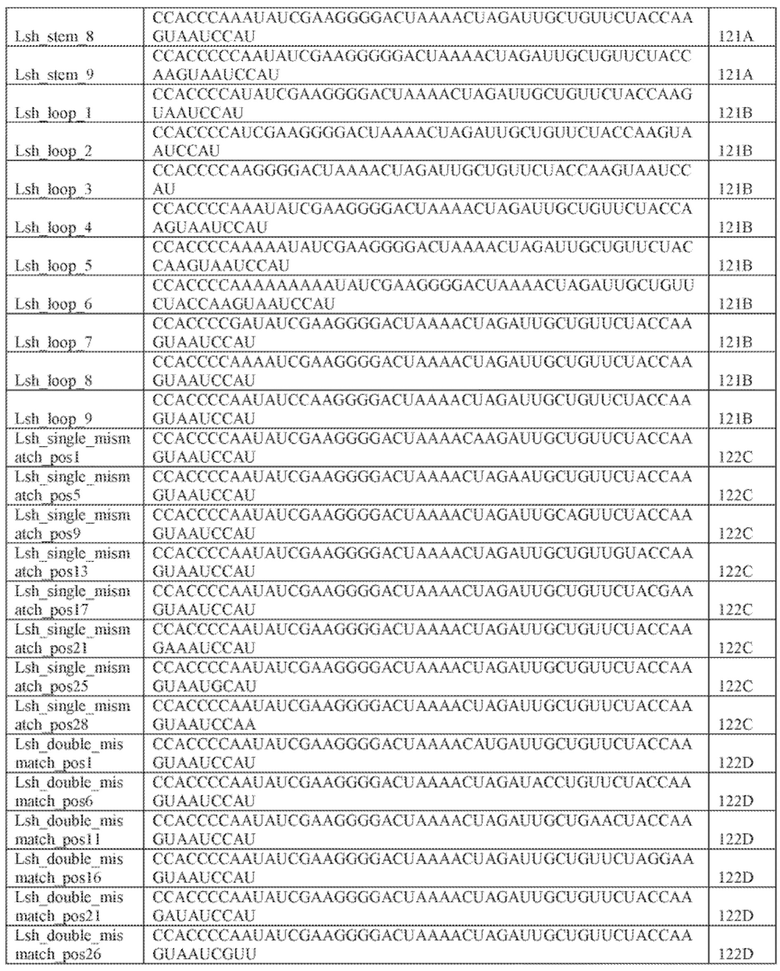
Таблица В. оцРНК-мишени, использованные в настоящем исследовании
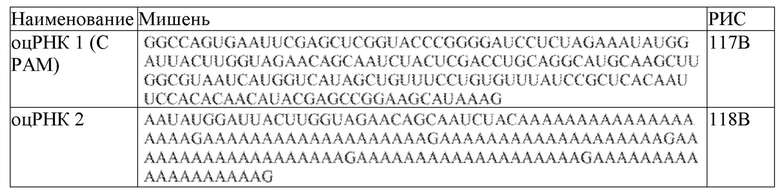
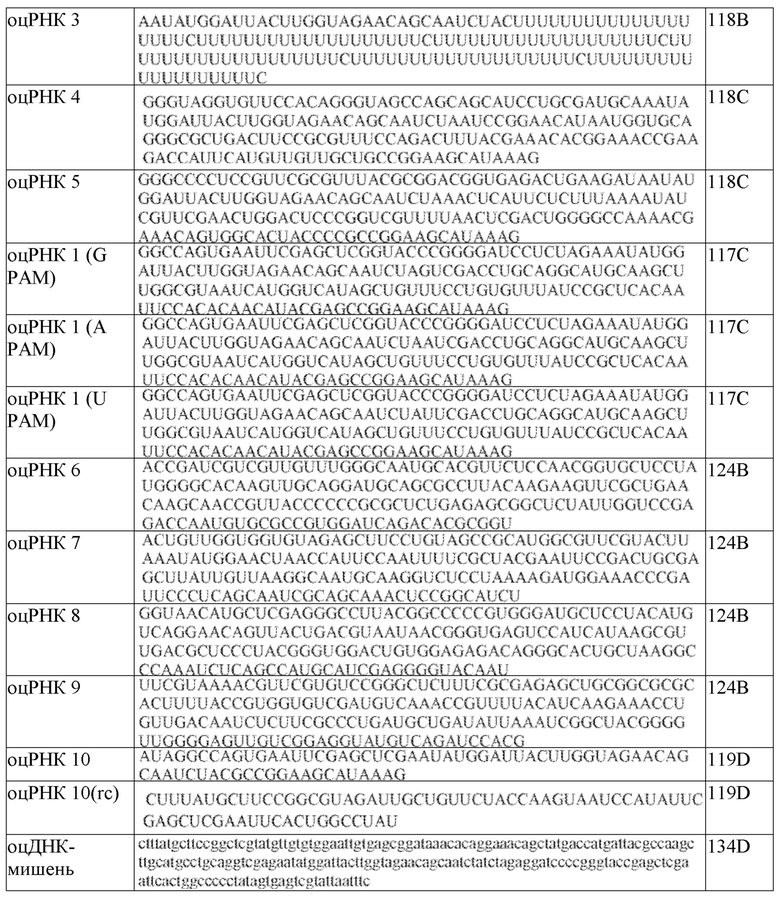
Таблица С. Спейсеры, использованные для экспериментов in vivo.
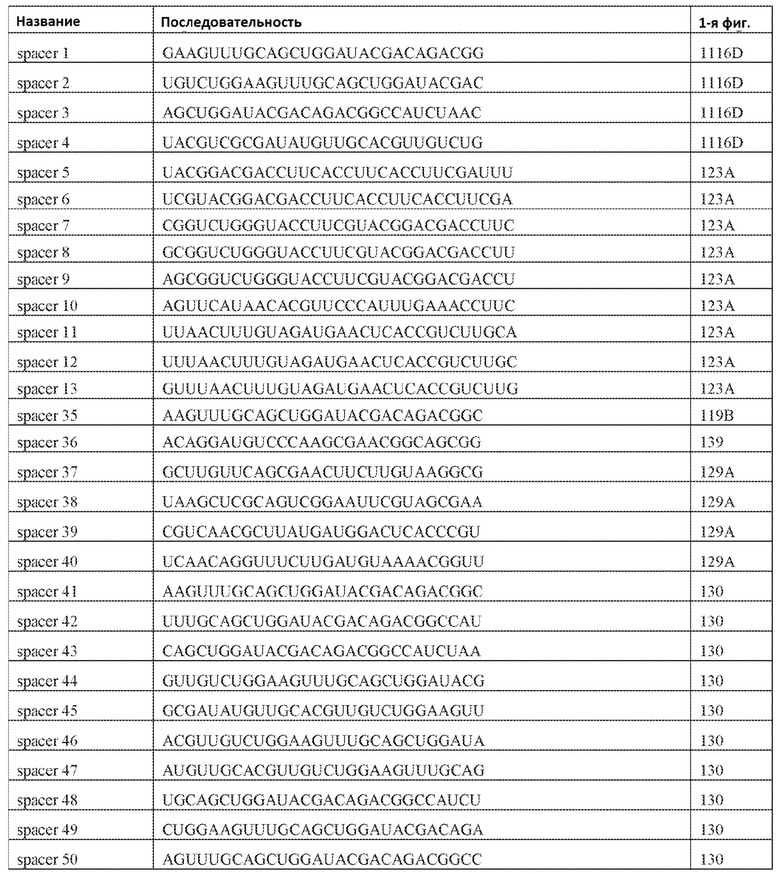
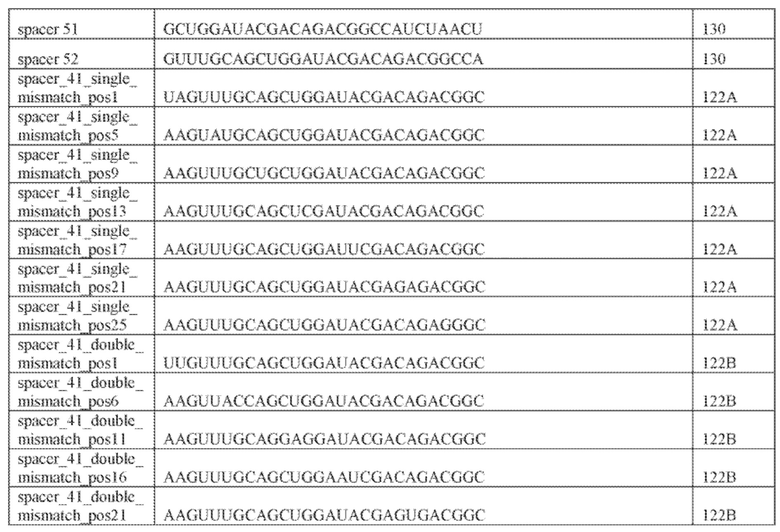
Гетерологическое реконструирование локуса L. shahii C2c2 в Escherichia coli подтверждает РНК-опосредованный иммунитет против РНК бактериофагов
[978] В качестве первого шага авторы изобретения исследовали, можно ли использовать LshC2c2 для обеспечения иммунитета к MS2 (G. Tamulaitis et al., Programmable RNA shredding by the type 111-A CRISPR-Cas system of Streptococcus thermophilus. Mol Cell 56, 506-517 (2014)), литическому одноцепочечному (ss) РНК-фагу, чей жизненный цикл проходит без образования ДНК-посредника и который легко заражает E. coli. Авторы изобретения сконструировали низкокопийную плазмиду, несущую весь локус LshC2c2 (pLshC2c2), чтобы обеспечить гетерологичное восстановление в E. coli (фиг. 126A). Учитывая, что экспрессированные зрелые cr-РНК из локуса LshC2c2 имеют максимальную длину спейсера 28 нуклеотидов (фиг. 126A) (S. Shmakov et al., Discovery and Functional Characterization of Diverse Class 2 CRISPR-Cas Systems. Mol. Cell 60, 385-397 (2015)), авторы изобретения синтезировали библиотеку из 3473 спейсерных последовательностей, выбирая все возможные сайты- мишени из 28 нуклеотидов в геноме фага MS2 и клонировали их как спейсеры в последовательность CRISPR pLshC2c2. После трансформации в E.coli клетки были инфицированы MS2 и далее были определены спейсерные последовательности в клетках, выживших в результате инфекции. Клетки, содержащие спейсеры, которые обеспечивают надежную защиту против MS2, будут пролиферировать быстрее, что приведет к обогащению этих спейсеров после роста в течение последующих 16 часов. Ряд спейсеров последовательно обогащался в двух независимых повторностях, что указывало на то, что они обеспечивали сильную защиту против MS2 (107 спейсеров показали> 1,3 log2-кратное обогащение в обеих репликах, фиг. 116B и ФИГ. 127A-B). Анализируя фланкирующие области протоспасера на геноме MS2, соответствующем 107 обогащенным спейсерам, авторы изобретения обнаружили, что спейсеры с G, непосредственно прилегающие к 3'-концу протоспейсера, работали хуже, чем в случае H (то есть A, U или С), что указывает на единый нуклеотидный PAM, H (фиг. 116C и фиг. 127C-D, 128).
[979] Для подтверждения интерференционной активности обогащенных спейсеров авторы изобретения индивидуально клонировали четыре самых обогащенных спейсера в pLshC2c2 последовательностях CRISPR и наблюдали 3-4-log-кратное уменьшение (103-104) образования бляшек в соответствии с уровнем обогащения, наблюдаемым при скрининге (фиг. 116B и фиг. 129). Чтобы подтвердить PAM, авторы изобретения клонировали шестнадцать направляющих молекул, нацеливающих на различные области гена mat фага MS2 (4 направляющих на один возможный однонуклеотидный PAM). Авторы изобретения обнаружили, что все 16 cr-РНК опосредуют интерференцию MS2, хотя более высокие уровни резистентности наблюдаются для С, A и U PAM-нацеливающих направляющих молекул (фиг. 116D, 116E и фиг,130), что указывает на то, что C2c2 можно эффективно перепрограммировать с помощью cr-РНК для нацеливания на сайты в геноме MS2.
C2c2 является одноэффекторной эндонуклеазой, которая опосредует расщепление оцРНК при помощи одной напрявляющей последовательности cr-РНК
[980] Чтобы проверить, опосредует ли LshC2c2 интерференцию фага, способствуя расщеплению оцРНК, направляемому cr-РНК, авторы изобретения очистили белок LshC2c2 (фиг. 131) и проанализировали его способность расщеплять in vitro мишень оцРНК длиной 173 нуклеотидов (фиг. 117A и фиг. 132), содержащую протоспейсер С-PAM (оцРНК-мишень 1 с протоспейсером 14). Ранее авторы изобретения обнаружили, что зрелые cr-РНК LshC2c2 содержат прямой повтор (DR) длиной 28 нуклеотидов и спейсер длиной 28 нуклеотидов (фиг. 126А) (S. Shmakov et al., Discovery and Functional Characterization of Diverse Class 2 CRISPR-Cas Systems. Mol Cell 60, 385-397 (2015)), и поэтому авторы изобретения сконструировали транскрибированную invitro cr-РНК со спейсером длинной 28 нуклеотидов, комплементарным протоспейсеру 14 на оцРНК-мишени 1. Авторы изобретения обнаружили, что LshC2c2 эффективно расщепляет оцРНК в условиях, зависящих от хелатируемого Mg2+- и cr-РНК (фиг. 117В и фиг. 133). Чтобы исследовать расщепление субстратов дцРНК, авторы изобретения произвели отжиг комплементарных олигонуклеотидов РНК в областях, фланкирующих сайт мишени cr-РНК. Этот частично двухцепочечный РНК-субстрат не был расщеплен LshC2c2, что указывает на его специфичность для оцРНК (фиг. 134A-B).
[981] Чтобы далее охарактеризовать ограничения, накладываемые последовательностью на расщепление РНК белком LshC2c2, авторы изобретения проверили дополнительные cr-РНК, комплементарные различным версиям оцРНК-мишени l, где протоспейсеру 14 предшествует каждый из вариантов PAM. Результаты этого эксперимента подтвердили предпочтение С, А и U PAM-последовательностей с небольшой активностью расщепления, обнаруженной для мишени с G PAM (фиг. 117C). Кроме того, авторы изобретения разработали 5 cr-РНК для каждого возможного PAM (всего 20) для оцРНК-мишени 1 и оценили ферментативную активность для LshC2c2 в паре с каждой из этих cr-РНК. Как и ожидалось, авторы изобретения обнаружили меньшую активность расщепления для G-PAM, нацеливающих cr-РНК, по сравнению с другими тестируемыми cr-РНК (фиг. 117D).
[982] LshC2c2 тестировали на активность по расщеплению ДНК in vitro. Авторы изобретения создали библиотеку дц ДНК-плазмид с протоспейсером 14, которому предшествовали 7 случайных нуклеотидов для учета любых требований к PAM. При инкубировании с белком LshC2c2 и в случае комплементарности cr-РНК протоспейсеру 14, никакого расщепления библиотеки плазмиды дцДНК не наблюдалось (фиг. 134C). Авторы изобретения также не наблюдали расщепления при нацеливании на оцДНК-версию оцРНК-мишени 1 (фиг. 134D). Дабы исключить совместное транскрипционное расщепление ДНК, которое наблюдалось в системах CRISPR-Cas типа III (P. Samai et al., Co-transcriptional DNA and RNA Cleavage during Type III CRISPR-Cas Immunity. Cell 161, 1164-1174 (2015)), авторы изобретения повторили анализ ко-транскрипционного расщепления РНК-полимеразы в E. coli (P. Samai et al., Co-transcriptional DNA and RNA Cleavage during Type III CRISPR-Cas Immunity. Cell 161, 1164-1174 (2015)) (фиг. 135A), экспрессирующий оцРНК-мишень из ДНК-субстрата. Используя этот способ анализа с очищенным LshC2c2 и cr-РНК, тартерирующую оцРНК-мишень 1, авторы изобретения все рано не наблюдали никакого расщепления ДНК (фиг. 135B). В целом, эти результаты показывают, что C2c2 расщепляет специфические сайты оцРНК, направленные комплементарностью к мишени, закодированной в cr-РНК, при 3' H PAM.
Ферментативная активность C2c2 зависит от последовательности-мишени и вторичной структуры
[983] Учитывая неэффективность C2c2 при расщеплении дцРНК и тот факт, что оцРНК образует сложные вторичные структуры, авторы изобретения полагали, что на расщепление C2c2 может влиять вторичная структура мишени оцРНК. При конструировании различных cr-РНК для всех возможных участков на оцРНК-мишени 1 (фиг. 117D) такая же картина расщепления наблюдалась независимо от положения cr-РНК вдоль РНК-мишени, что указывало на то, что зависимое от cr-РНК расщепление определялось некоторыми признаками последовательности-мишени, а не расстоянием от места взаимодействия. Авторы изобретения предположили, что комплекс LshC2c2-cr-РНК связывает мишень и расщепляет открытые области вторичных структурных элементов оцРНК, демонстрируя потенциальное предпочтение для некоторых нуклеотидов. Авторы изобретения проанализировали эффективность расщепления мишеней гомополимерной РНК (протоспейсер длиной 28 нуклеотидов, увеличенный при помощи 120 нуклеотидов A или U, регулярно перемежающихся с одиночными основаниями G или C, чтобы обеспечить синтез олигонуклеотида, и обнаружили, что LshC2c2 предпочтительно расщепляет мишень урацила по сравнению с аденином (фиг. 118А-В). Чтобы оценить влияние РНК-мишени на картину расщепления, авторы изобретения протестировали расщепление трех мишеней оцРНК с различными последовательностями, фланкирующими постоянный протоспейсер длиной 28 нуклеотидов, и обнаружили три различных типа расщепления (фиг. 118C). Секвенирование РНК продуктов расщепления для трех мишеней показало, что сайты расщепления в основном локализованы в богатых урацилом областях оцРНК или участках соединения оцРНК-дцДНК внутри предсказанных in silico участков со-фолдинга последовательности-мишени при помощи cr-РНК (фиг. 118D-I).
HEPN-домены C2c2 опосредуют РНК-направляемое расщепление оц-РНК
[984] Предшествующий анализ C2c2 проведенный способами биоинформатики предположил, что домены HEPN потенциально ответственны за наблюдаемую нами каталитическую активность (S. Shmakov et al., Discovery and Functional Characterization of Diverse Class 2 CRISPR-Cas Systems. Mol Cell 60, 385-397 (2015)). Каждый из двух доменов HEPN эффектора C2c2 содержит диаду консервативных остатков аргинина и гистидина (фиг. 119A), что согласуется с каталитическим механизмом HEPN эндо-РНК-азы endoRNAse (V. Anantharaman, K. S. Makarova, A. M. Burroughs, E. V. Koonin, L. Aravind, Comprehensive analysis of the HEPN superfamily: identification of novel roles in intra-genomic conflicts, defense, pathogenesis and RNA processing. Biol Direct 8, 15 (2013); O. Niewoehner, M. Jinek, Structural basis for the endoribonuclease activity of the type III-A CRISPR-associated protein Csm6. RNA 22, 318-329 (2016); N. F. Sheppard, С. V. Glover, 3rd, R. M. Terns, M. P. Terns, The CRISPR-associated Csx1 protein of Pyrococcus furiosus is an adenosine-specific endoribonuclease. RNA 22, 216-224 (2016)). Чтобы проверить, необходимы ли эти предсказанные каталитические остатки для обеднения оцРНК in vivo, путем внесения мутаций авторы изобретения отдельно заменили каждый участок на аланин (R597A, H602A, R1278A, H1283A) в локусных плазмидах LshC2c2 и анализировали на наличие интерференции MS2. Ни одна из четырех мутантных плазмид не могла защитить E.coli от проникновения фага в клетку (фиг. 119B и фиг. 136).
[985] Для подтверждения этих результатов in vitro четыре мутантных белка c точечной мутацией были очищены и протестированы на наличие способности расщеплять 5'-концевую меченую мишень оцРНК 1 (фиг. 119C). Полностью подтверждая результаты, полученные авторами изобретения in vivo, все четыре мутации отменяли реакцию расщепления. Неспособность любого из двух доменов HEPN дикого типа компенсировать инактивацию другого подразумевает совместную работу двух доменов, что согласуется с наблюдениями, что в качестве димеров функционируют несколько бактериальных и эукариотических одиночных HEPN-белков (O. Niewoehner, M. Jinek, Structural basis for the endoribonuclease activity of the type Ш-А CRISPR-associated protein Csm6. RNA. 22, 318-329 (2016); N. F. Sheppard, С. V. Glover, 3rd, R. M. Terns, M. P. Terns, The CRISPR- associated Csx1 protein of Pyrococcus furiosus is an adenosine-specific endoribonuclease, RNA 22, 216-224 (2016); G. Kozlov et al., Structural Basis of Defects in the Sacsin HEPN Domain Responsible for Autosomal Recessive Spastic Ataxia of Charlevoix-Saguenay (ARSACS). I Biol Chem 286, 20407-20412 (2011)).
[986] Каталитически неактивные варианты Cas9 сохраняют способность к связыванию с ДНК-мишенью, что позволяет создавать программируемые ДНК-связывающие белки (G. Gasilinas, R. Barrangou, P. Horvath, V, Siksnys, Cas9-cr-РНК ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci U S A 109, E2579-2586 (2012); M. Jinek et al., A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816-821 (2012)). Для определения того, являются ли связывание с мишенью и активность расщепления LshC2c2 таким же образом разделяемыми, анализы сдвига электрофоретической подвижности (EMSA) были выполнены как на диком типе (фиг. 119D), так и с R1278A-мутантом LshC2c2 (фиг. 119E) в комплексе с cr-РНК. Комплекс LshC2c2 дикого типа демонстрировал сильное связывание (KD ~46 нМ, фиг. 137A), в частности, с мишенью 10 оцРНК, но не с не-мишенью оцРНК (обратно комплементарной мишени 10 оцРНК). У мутантного C2c2 комплекса R1278A обнаружено еще более сильное (KD ~7 нМ, фиг. , 137B) специфическое связывание, что указывает на то, что эта мутация домена HEPN приводит к каталитически неактивному РНК-программируемому РНК-связывающему белку. Только белок LshC2c2 или только cr-РНК по отдельности показали существенно уменьшенные уровни сродства к мишени, как и ожидалось (фиг. 137C-E).
[987] Эти результаты демонстрируют, что C2c2 расщепляет РНК с использованием каталитического механизма, отличного от других известных CRSSPR-ассоциированных РНК-аз. В частности, мультибелковые комплексы Csm и Cmr типа III основаны на кислых остатках каталитических доменов RRM, тогда как C2c2 расщепляет РНК с помощью консервативныхи основных остатков двух его доменов HEPN.
Структурные требования и необходимые последовательности для cr-РНК C2c2
[988] Подобно системам типа V-B (Cpf1) (B. Zetsche et al., Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell 163, 759-771 (2015) cr-РНК LshC2c2 содержит единственную шпильку в прямом повторе (DR), поэтому можно предположить, что вторичная структура cr-РНК облегчает взаимодействие с LshC2c2. Для изучения такой возможности, авторы изобретения сначала исследовали требования к длине спейсерной последовательности для расщепления оцРНК и обнаружили, что LshC2c2 требует спейсеров длиной не менее 22 нуклеотидов для эффективного расщепления мишени 1 оцРНК (фиг. 120A). Авторы изобретения также обнаружили, что структура шпильки cr-РНК имеет определяющее значение для расщепления оцРНК, поскольку укорочение прямых повторов (DR) нарушало структуру шпильки, а следовательно, и отменяло расщепление мишени (фиг. 120B). Таким образом, требуется DR более 24 нуклеотидов длиной для поддержания структуры шпильки, необходимой LshC2c2 для опосредованного расщепления оцРНК.
[989] Затем авторы изобретения изучили влияние модификаций в стебле и петле прямых повторов cr-РНК на активность расщепления. Одиночные инверсии в парах оснований стебля, при которых он сохранял свою структуру, не влияли на активность комплекса LshC2c2, но инвертирования всех четырех пар G-C в стебле прекращало расщепление, несмотря на сохранение структуры дуплекса (фиг. 121 А). Другие видоизменения, которые привносили изломы и уменьшали или увеличивали спаривание оснований в стебле, также приводили к полному прекращению или значительному подавлению расщепления, делая возможным предположение, что длина стебля cr-РНК важна для образования комплекса (с LshC2c2) и активности расщепления (фиг. 121 A). Посредством ряда модификаций авторы изобретения обнаружили, что удаление петель прекращает процесс расщепления, тогда как вставки и замены в основном поддерживают некоторый уровень активности расщепления (фиг. 121B). В целом эти результаты показывают, что LshC2c2 распознает структурные характеристики родственной ему cr-РНК, но допускает возможность как вставки петель, так и других протестированных замен оснований. Эти результаты имеют значение для дальнейшей разработки и применения инструментов на основе C2c2, для которых требуется разработка направляющих (guide) молекул для привлечения эффекторов или модулирования активности (S. Kiani et al., Cas9 gRNA engineering for genome editing, activation and repression. Nat Methods 12, 1051-1054 (2015); S. Konermann et al., Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature 517, 583-588 (2015); J. E. Dahlman et al., Orthogonal gene knockout and activation with a cataiytically active Cas9 nuclease. Nat Biotechnol 33, 1159-1161 (2015)).
Расщепление при помощи C2c2 чувствительно к двойным нарушениям комплементарности в дуплексе cr-РНК-мишени
[990] Авторы изобретения проверили чувствительность системы LshC2c2 на возможность продолжать активность при единичных нарушениях комплементарности между направляющей cr-РНК и РНК-мишенью путем мутации отдельных оснований на всей длине спейсера соответствующими изменениями комплементарных оснований (например, A на U) и количественного образования бляшек с этими некомплементарными спейсерами при заражении фагом MS2 (фиг. 122A и фиг. 138). Авторы изобретения обнаружили, что C2c2 полностью толерантен к единичным нарушениям комплементарности по длине спейсера, поскольку такие некомплементарные спейсеры препятствуют распространению фага с такой же эффективностью, как и полностью комплементарные спейсеры. Однако, когда авторы изобретения вводили последовательные двойные замены в спейсер, авторы изобретения обнаружили сокращение эффективности защиты в 1000 раз ~при некомплементарности в центре, но не на 5'- или 3'-конце, cr-РНК (фиг. 122B и фиг. 138). Это наблюдение указывает на наличие чувствительной к рассогласованию в области затравки в центре дуплекса cr-РНК-мишени.
[991] Авторы изобретения также оценили необходимые LshC2c2 условия для связывания направляющей и последовательности-мишени in vitro. С этой целью авторы изобретения создали множество транскрибируемых in vitro cr-РНК с нарушениями комплементарности, расположенными одинаково на всей длине спейсера. При проведении инкубации с белком LshC2c2 все единичные нарушения комплементарности в cr-РНК не приводили к нарушениям расщепления (фиг. 122C), что согласуется с результатами, полученными авторами изобретения in vivo. При тестировании при помощи набора последовательных двойных мутантных cr-РНКs, LshC2c2 был неспособен расщеплять РНК-мишень, в случае локализации нарушений в центре, но не на 5'- или 3'-конце cr-РНК (фиг. 122D), что подтверждает существование сердцевинной области-затравки.
[992] Была также оценена чувствительность системы LshC2c2 к двойным и тройным нарушениям комплементаности. Двойные несоответствия были локализованы в разных местах (фиг. 143A), тогда как тройные несоответствия были расположены последовательно (фиг. 143B). Было показано, что чувствительность расщепления зависит от расположения нарушения комплементарности. Нарушения комплементарности, близкие к области прямых повторов (DR), не способствовали расщеплению мишени, тогда как дистальные приводили к обнаружимому расщеплению.
[993] Система LshC2c2 также чувствительна к нарушениям комплементарности и делециям в области прямого повтора. Единичные нарушения комплементарности и одиночные делеции оснований, как правило, достаточны, чтобы нарушить расщепление оцРНК. Только одно нарушение комплементарности (мутант 7) все же позволяло сохранить низкий уровень расщепления (фиг. 144).
C2c2 может быть перепрограммирован для опосредования специфичного нокдауна мРНК in vivo
[994] Учитывая способность C2c2 расщеплять оцРНК-мишень специфичным к последовательности cr-РНК образом, авторы изобретения проверили, можно ли перепрограммировать LshC2c2 для деградации выбранных нефаговых мишеней оцРНК и, в частности, мРНК in vivo. С этой целью авторы изобретения одновременно трансформировали E. coli с помощью плазмиды, кодирующей LshC2c2, и cr-РНК, нацеливающей на мРНК красного флуоресцентного белка (RFP), а также совместимой плазмиды, экспрессирующей RFP (фиг. 123A). Авторы изобретения наблюдали уменьшение RFP-положительных клеток примерно на 20% до 92% для cr-РНК, нацеливающей на протоспейсеры, фланкированные С, A или U-PAM для образцов, совпадающих по оптической плотности (фиг. 123B, C). В качестве контроля авторы изобретения тестировали cr-РНК, содержащие обратно комплементарные последовательности (нацеливающие на дцДНК-плазмиду) к наиболее эффективным нацеленным на мРНК спейсерам RFP. Как и ожидалось, авторы изобретения не наблюдали снижения флуоресценции RFP при воздействии cr-РНК (фиг. 123B). Авторы изобретения также подтвердили, что мутация каталитических остатков аргинина в домене HEPN до аланина исключает нокдаун RFP (фиг. 139). Таким образом, C2c2 способен к общему перенацеливанию на произвольные субстраты оцРНК, регулируемые только предсказуемыми взаимодействиями нуклеиновых кислот.
[995] Когда авторы изобретения исследовали скорость роста клеток, имеющих спейсер, нацеливающий на RFP, с наибольшим уровнем нокдауна RFP, авторы изобретения заметили, что скорость значительно уменьшилась (фиг. 123 A, спейсер 7). Чтобы определить причину этого ограничения роста, авторы изобретения исследовали, было ли влияние на рост опосредовано активностью LshC2c2 по нацеливанию на мРНК RFP путем введения плазмиды, индуцируемой RFP, и с локуса LshC2c2, нацеленного на RFP, в E.coli. Используя эту систему, авторы изобретения обнаружили, что при индукции транскрипции RFP клетки с нокаутом RFP показали существенное подавление роста, которое не наблюдалось при контрольных экспериментах без нацеливания (фиг. 123D, E). Однако при отсутствии транскрипции RFP авторы изобретения не наблюдали ограничения роста, так же, как не наблюдали зависимого от транскрипции нацеливания на ДНК биохимическом эксперименте (фиг. 135), что позволяет предположить, что нацеливание на РНК, вероятно, является основным фактором появления этого фенотипа с ограничением роста. Не ограничиваясь только теорией, одним из возможных объяснений этого эффекта является то, что системы CRISPR с эффекторным белком C2c2 могут функционировать для предотвращения размножения вируса, неспецифично расщепляя клеточные мРНК и индуцируя снижение клеточного деления, запрограммированную гибель клеток (PCD) или состояние покоя ((K. S. Makarova, Y. I. Wolf, E. V. Koonin, Comprehensive comparative-genomic analysis of type 2 toxin-antitoxin systems and related mobile stress response systems in prokaryotes. Biol Direct 4, 19 (2009); F. Hayes, L. Van Melderen, Toxins-antitoxins: diversity, evolution and function. Crit Rev Biochem Mol Biol 46, 386-408 (2011)).
C2c2 расщепляет побочную РНК в дополнение к оцРНК, на которую нацелена cr-РНК
[996] По сравнению с Cas9 и Cpf1, которые расщепляют ДНК внутри гетеродуплекса мишени cr-РНК в определенном положении, возвращаясь в неактивное состояние после расщепления, C2c2 расщепляет РНК-мишень вне сайта связывания cr-РНК на разных расстояниях в зависимости от фланкирующей последовательности, предположительно в пределах выступающих петель оцРНК (фиг. 118D-I). Эта наблюдаемая гибкость в расщеплении позволяет нам рассмотреть возможность расщепления соседних нецелевых оцРНК при связывании и активации мишеней C2c2. Соответственно, C2c2 может вызывать PCD через механизм из двух составляющих: стадию прайминга, в которой комплексы C2c2-cr-РНК связываются с целевыми сайтами и расщепляют оцРНК, направляемые cr-РНК, на второй стадии, в которой праймированный C2c2 расщепляет нецелевую, побочную РНК неспецифически. Чтобы проверить эту гипотезу, авторы изобретения проводили реакции расщепления in vitro, которые включали, помимо LshC2c2, cr-РНК и ее РНК-мишень, одну из четырех несвязанных с ними молекул РНК без какой-либо комплементарностью к направляющей cr-РНК (фиг. 124A). Эти эксперименты показали, что, в то время как комплекс LshC2c2-cr-РНК не опосредовал расщепление любой из четырех коллатеральных (побочных) РНК в отсутствие целевой РНК, все четыре были эффективно деградированы в присутствии целевой РНК (фиг. 124B и фиг. 140А). Кроме того, мутанты R597A и R1278A по домену HEPN не смогли отщеплять побочную РНК (фиг. 140B). Эти результаты указывают на HEPN-зависимый механизм, посредством которого C2c2 в комплексе с cr-РНК активируется при связывании с целевой РНК и затем расщепляет любые ближайшие оцРНК-мишени. Такое неизбирательное расщепление РНК может вызывать цитотоксичность, что приводит к наблюдаемому ингибированию роста. Эти данные указывают на то, что в дополнение к их роли в прямом подавлении РНК-вирусов системы CRISPR-Cas типа VI могут функционировать в качестве медиаторов различного рода индукции программируемой клеточной гибели (PCD)/покоящегося состояния, которые специфически инициируются распознаваемыми чужеродными геномами (фиг. 125). Согласно этому сценарию, состояние покоя замедляло бы инфекцию и предоставляло бы дополнительное время адаптивному иммунитету для подавления инфекции; в случае неуспеха адаптивного иммунитета, суицидальная роль C2c2 будет преобладать, и распространение инфекции будет ограничено. Такой механизм входит в ранее предложенную схему связи между адаптивным иммунитетом и программируемой клеточной гибелью (PCD) во время защитной реакции CRISPR-Cas (K. S. Makarova, V. Anantharaman, L. Aravind, E. V. Koonin, Live virus-free or die: coupling of antivirus immunity and programmed suicide or dormancy in prokaryotes. Biol Direct 7, 40 (2012)).
Пример 8: Экспрессия C2c2 в эукариотических клетках
[997] Ряд ортологов C2c2 были кодон-оптимизированы для экспрессии в клетках млекопитающих с использованием вектора экспрессии млекопитающих. Различные ортологи C2c2 были трансфицированы в клетки HEK293T, и клеточную локализацию оценивали на основе экспрессии mCerry. Наблюдалась локализация цитоплазмы, а также ядерная локализация белка C2c2.
Пример 9: Активность C2c2 в эукариотических клетках
[998]. Анализ нацеливания на люциферазу проводили с различными гРНК, направленными против белка C2c2. Наблюдался эффективный нокдаун.
[999] Анализ нацеливания на основе экспрессии GFP выполнялся с помощью гРНК, нацеленных против EGFP. Экспрессию GFP определяли и сравнивали с ненацеливающей РНК (NT). Здесь тоже наблюдался эффективный нокдаун.
[1000] Анализ нацеливания проводили на разных эндогенных генах-мишенях в клетках HEK293 вместе с гРНК, направленными против эндогенных генов-мишеней C2c2. Определяли экспрессию белка соответствующих генов-мишеней (по сравнению с ненацеливающей (NT) гРНК). Наблюдался эффективный нокдаун различных генов-мишеней.
Методология для раздела примеры
Клонирование локуса С2с2 и библиотеки скрининга
[1001] Геномная ДНК из Leptotrichia shahii DSM 19757 (ATCC) была экстрагирована при помощи набора Blood & Cell Culture DNA Mini Kit (Qiagen), а локус С2с2 CRISPR был амплифицирован ПЦР и клонирован в остов pACYC184 с устойчивостью к хлорамфениколу. Для перенацеливания на локус фага MS2 или эндогенные мишени спейсеры дикого типа удаляли из последовательности или заменяли посадочной площадкой ECO31I, дополненной спейсером и вырожденным повтором, совместимым с клонированием Golden Gate.
[1002] Пользовательская библиотека, состоящая из всех возможных спейсеров, нацеленных на геном бактериофага MS2, за исключением спейсеров, содержащих сайт рестрикции Eco31I, была синтезирована при помощи Twist Biosciences, клонированной в остов для перенацеливания клонированием Golden Gate, трансформированную в электрокомпетентные клетки Endura Duo (Lucigen) и затем очищенной с использованием NucleoBond XtraMaxiPrep EF (Machery-Nagel).
Бактериальный анализ интерференции
[1003] Для фагового скрининга 50 нг библиотеки плазмид трансформировали в компетентные клетки NovaBlue(DE3) (EMD Millipore) с последующим разрастания при 37°C в течение 30 минут. Затем клетки выращивали в Luria broth (LB), дополненном 25 мкг/мл хлорамфениколом (Sigma) в объеме 4,5 мл. Условия для культивирования фага обрабатывали 7×1010 б.о.е. бактериофага MS2 (ATCC). После 3 часов инкубирования при встряхивании при 37°C образцы наносили на чашки с агаром Луриа-Бертани (LB-agar), дополненные хлорамфениколом, и отбирали через 16 часов. ДНК была выделена с использованием NucleoBond Xtra Maxi Prep EF (Machery-Nagel), амплифицирована ПЦР и секвенирована с использованием MiSeq (Illumina) с набором парных концов на 150 циклов.
[1004] Для определения обогащенных спейсеров, спейсерные области были извлечены, подсчитаны и нормализованы на общее число считываний для каждого образца. Для данного PAM обогащение измеряли, как логарифмическое отношение в сравнении с исходной библиотекой с корректировкой псевдоотсчета 0,01. PAM выше порога обогащения 1,3, которые встречались в обоих биологических повторениях, были использованы для создания логотипов последовательностей (G. E. Crooks, G. Hon, J. M. Chandonia, S. E. Brenner, Web Logo: a sequence logo generator. Genome research 14, 1188- 1190 (2004)).
[1005] Для тестирования отдельных спейсеров на интерференцию фага MS2 олигонуклеотиды были заказаны из IDT, был произведен их отжиг, фосфорилирование полинуклеотид-киназой (New England Biosciences) и клонирование в остов локуса с помощью клонирования Golden Gate. Плазмиды трансформировали в штамм E.coli C3000, который был сделан компетентным с помощью набора Mix and Go kit (Zymo Research). Клетки С3000 высевались из культуры, полученной в течение ночи до OD600 равного 2, после чего были разведены 1:13 в Top Agar и помещены на чашки с LB-хлорамфениколом. Разведения фага MS2 были нанесены на чашки с помощью многоканальной пипетки и образование бляшек было зарегистрировано после инкубирования в течение ночи.
Анализ нацеливания на RFP
[1006] Устойчивая к ампициллину RFP-экспрессирующая плазмида (pRFP) была трансформирована в клетки DH5-alpha (New England Biolabs). Клетки, содержащие pRFP, затем были сделаны химически компетентными (Zymo Research Mix и Go) для использования в экспериментах по нацеливанию в нисходящем направлении с pLshC2c2. Спейсеры, нацеливающие на мРНК RFP, были клонированы в pLshC2c2, и эти плазмиды были трансформированы в химически компетентные клетки DH5-alpha pRFP. Затем клетки культивировали в течение ночи при двойном отборе в LB и подвергали анализу с помощью проточной цитометрии, когда они достигли OD600 4,0. Эффективность нокдауна была определена количественно как процент RFP-положительных клеток по сравнению с ненацеливающим спейсером в контроле (эндогенный локус LshC2c2 в pACYC184).
[1008] Для определения влияния активности LshC2c2 на рост клеток, авторы изобретения создали TetR-индуцируемую версию плазмиды RFP в pBR322 (pBR322_RFP). Авторы изобретения трансформировали клетки E.coli с этим вектором и затем сделали их химически компетентными (Zymo Research Mix and Go), чтобы подготовить к дальнейшим экспериментам. Авторы изобретения клонировали плазмиды pLshC2c2 с различными спейсерами, нацеленными на мРНК RFP, а также обратно комплементарными им контрольными спейсерами и трансформировали их в E.coli, несущую pBR322_RFP и просеивали их на чашки двойного отбора для поддержания обеих плазмид. Колонии затем собирали и культивировали в течение ночи в LB с двойным отбором. Бактерии разводили до OD600 0,1 и выращивали при температуре 37°С в течение часа с отбором только на устойчивость к хлорамфениколу. Затем экспрессию RFP вызывали использованием 350 нг/мл ангидротетрациклина, а измерения OD проводили каждые 5 минут при непрерывном встряхивании в ридере микропланшетов BioTek Synergy 2.
Получение нуклеиновой кислоты С2с2
[1009] Кодон-оптимизированный для экспрессии в млекопитающих ген С2с2 (Leptotrichia shahii) был синтезирован и клонирован в бактериальную экспрессионную плазмиду. Клетки E.coli (BL21(DE3)) были трансформированы и культивированы в течение одной ночи при температуре 37°С. Белок был очищен при помощи гистидиновых меток и аффинной хроматографии с колонками с Ni-NTA и затем дополнительно очищен при помощи гель-фильтрации FPLC.
[1010] Матрицы нуклеиновой кислоты для транскрипции Т7 были синтезированы из IDT. Был произведен отжиг матриц cr-РНК до короткого праймера Т7 и инкубация с полимеразой Т7 в течение ночи при температуре 30°С.
[1011] 5'-концевое мечение была выполнена с использованием 5'-олигонуклеотидного набора (VectorLabs) и с зондом maleimide-IR800 (Licor). 3'-концевое мечение было выполнено с использованием 3'-олигонуклеотидного набора (Sigma) с использованием ddUTP-Cy5. Маркированные зонды были очищены с использованием колонок Clean and Concentrator (Zymo).
Очистка белка С2с2
[1012] Кодон-оптимизированный для экспрессии в млекопитающих ген C2c2 (Leptotrichia shahii) был синтезирован (GenScript) и клонирован в бактериальный экспрессирующий вектор (6-His-MBP-TEV-Cpf1, вектор на основе pET, любезно предоставленный нам Doug Daniels). 12 литров культуральной среды Terrific Broth growth с 100 мкг/мл ампициллина инокулировали с 10 мл культуры One Shot® BL21(DE3)pLysE (Invitrogen), содержащей конструкцию для экспрессии LshC2c2, на протяжении ночи. Культуральную среду и инокулянт выращивали при температуре 37°С до тех пор, пока плотность клеток не достигала OD600 0,2, затем температуру снижали до 21°С. Рост клеток продолжался до достижения OD600 0,6, когда была добавлена конечная концентрация 500 мкМ IPTG для индуцирования экспрессии MBP-C2c2. Культуру индуцировали в течение 14-18 часов перед отбором клеток и замораживанием до -80°С перед очисткой.
[1013] Клеточную массу ресуспендировали в 200 мл буфера для лизиса (50 мкМ Hepes pH 7, 2M NaCl, 5 мМ MgCl2, 20 мМ имидазола), дополненного ингибиторами протеазы (Roche cOmplete, свободный от EDTA) и лизоцимом. После гомогенизации клетки лизировали ультразвуком (Branson Sonifier 450), затем центрифугировали при 10000g в течение 1 часа, чтобы очистить лизат. Лизат фильтровали через 0,22-микронные фильтры (Millipore, Steric-up), нанесенные на Ni-NTA-сверхчистую никелевую смолу (Qiagen), промывали, а затем элюировали в градиенте имидазола. Фракции, содержащие белок ожидаемого размера, объединяли, добавляли протеазу TEV (Sigma) и образец диализировали в течение одной ночи в TEV-буфере (500 мкМ NaCl, 50 мМ Hepes pH 7,5 мМ MgCl, 2 мМ DTT). После диализа расщепление TEV подтверждали SDS-PAGE и образец концентрировали до 500 мкл перед загрузкой на гель-фильтрационную колонку (HiLoad 16/600 Superdex 200) через FPLC (АКТА Pure). Фракции гель-фильтрации анализировали с помощью SDS-PAGE; фракции, содержащие C2c2, объединяли и концентрировали до 200 мкл и либо использовали непосредственно для биохимических анализов, либо замораживали при -80°C для хранения. Стандарты для гель-фильтрации использовались на той же колонке, уравновешенной в 2 М NaCl, Hepes pH 7,0 для расчета приблизительного размера LshC2c2.
Подготовка нуклеиновой кислоты-мишени
[1014] ДНК-олиго-матрицы для транскрипции T7 были заказаны из IDT. Матрицы для cr-РНК отжигали на коротком праймере T7 и инкубировали с T7-полимеразой в течение ночи при 30°C с использованием набора для синтеза РНК HiScribe T7 Quick High Yield RNA Synthesis (New England Biolabs). Матрицы-мишени были амплифицировали ПЦР с получением дцДНК и затем инкубированы с T7-полимеразой при 30°C в течение ночи с использованием того же набора.
[1015] 5'-концевое мечение было проведено с использованием 5'-олигонуклеотидного набора (VectorLabs) и с зондом maleimide-IR80Q (Licor). 3'-концевое мечение было проведено с использованием 3'-олигонуклеотидного набора для маркировки (Sigma) с использованием ddUTP-Cy5. Маркированные зонды очищали с использованием колонок Clean и Concentrator (Zymo Research).
Способы исследования нуклеазы
[1016]. Анализ нуклеазы проводили с 160 нМ меченной на конце оцРНК-мишени. 200 нМ очищенного LshC2c2 и 100 нМ cr-РНК, если не указано иначе, в буфере для анализа нуклеазы (40 мМ Трис-HCl, 60 мМ NaCl, 6 мМ MgCl2, pH 7,3). Реакции проводили в течение 1 часа при 37°C (если не указано иное), а затем останавливали протеиназой К и ЭДТА в течение 15 минут при 37°С. Реакции затем денатурировали денатрурирующим буфер 6 М мочевины при 95°С в течение 5 минут. Образцы анализировали с помощью гель-электрофореза с 10% PAGE и TBE-мочевиной при 45°C. Гели были визуализированы с помощью сканера Licor Odyssey.
Анализ сдвига электрофоретической подвижности
[1017] Эксперименты по связыванию оцРНК-мишени выполнялись с помощью серии полулогарифмических разведений (cr-РНК и LshC2c2) с концентрациями от 2 мкМ до 0,2 пМ (или от 1 мкМ до 0,1 пМ в случае LshC2c2 R1278A). Анализ связывания проводили в буфере для определения нуклеазы, дополненном 10 мМ ЭДТА, для предотвращения разрезания, 5% глицерина и 10 мкг/мл гепарина во избежание неспецифических взаимодействий комплекса с РНК-мишенью. Реакции инкубировали при 37°С в течение 20 минут, затем они были разрешены на 6% гелях PAGE TBE при 4°C (используя буфер 0,5X TBE). Гели были визуализированы с помощью сканера Licor Odyssey.
NGS in vitro расщепленной РНК
[1018] Способы на определение нуклеазы in vitro проводили, как описано выше, с использованием немаркированных мишеней оцРНК. Через час реакцию останавливали при помощи протеиназы К+ЭДТА, а затем очищали в колонке (Zymo Clean с концентратором). Образцы РНК были затем обработаны PNK и 5'-полифосфатазой (Epicenter) перед подготовкой библиотеки для NGS с использованием NEBNext Small RNA Library Prep Set для секвенирования с помощью Illumina. Библиотеки были секвенированы на Illumina MiSeq с достаточной глубиной и проанализированы с использованием инструмента для выравнивания BWA (H. Li, R. Durbin, Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics 25, 1754-1760 (2009)).
Анализ расщепления ко-транскрипционной ДНК in vitro
[1019] Анализ котранскрипционного расщепления ДНК комплексом RNAP в E. coli, проводился так, как описано ранее (Samai et al., Cell, 2015). В кратком изложении, для 0,8 пмоль цепи оцДНК-матрицы проводили отжиг 1,6 пмоль РНК в буфере транскрипции (из центральной части фермента RNAP E. coli New England Biolabs) без магния для предотвращения гидролиза РНК. Реакцию проводили с 0,75 мкл коровой части фермента RNAP E. coli и магния, инкубировали при 25°C в течение 3 минут, а затем переносили на 37°C. Добавляли 1 пмоль свежеденатурированной некодирующей цепи (NTS) и инкубировали при 37°C в течение 15 минут для получения элонгационных комплексов (EC). К EC добавляли 4 пмоль комплексов LshC2C2-cr-РНК вместе с 1,25 мМ RNTP, и транскрипции давали возможность протекать 1 ч при 37°C. ДНК была разведена в 10%-гелях PAGE TBE-Urea после обработки РНК-азой и протеиназой К.
[1020] Хотя предпочтительные варианты осуществления настоящего изобретения были продемонстрированы и описаны в настоящем описании, специалистам в данной области будет очевидно, что такие варианты осуществления предоставляются только в качестве примера. Многочисленные вариации, изменения и замены теперь могут быть разработаны специалистами в данной области без отступления от изобретения. Следует понимать, что различные альтернативы вариантам осуществления изобретения, описанным в настоящем описании, могут быть использованы при осуществлении изобретения. Предполагается, что нижеследующая формула изобретения определяет объем изобретения и что способы и структуры, входящие в объем этих притязаний и их эквиваленты, будут охвачены таким образом.
--->
SEQUENCE LISTING
<110> The Broad Institute, Inc.
The United States of America, as represented by the
Secretary, Department of Health and Human Services
Massachusetts Institute of Technology
President and Fellows of Harvard College
Rutgers, The State University of New Jersey
Skolkovo Institute of Science and Technology
Severinov, Konstantin
Zhang, Feng
Wolf, Yuri I.
Shmakov, Sergey
Semenova, Ekaterina
Minakhin, Leonid
Makarova, Kira S.
Koonin, Eugene
Konermann, Silvana
Joung, Julia
Gootenberg, Jonathan S.
Abudayyeh, Omar O.
Lander, Eric S.
<120> NOVEL CRISPR ENZYMES AND SYSTEMS
<130> BROD-3480US1
<140> US 15/482,603
<141> 2017-04-07
<150> PCT/US2016/038258
<151> 2016-06-17
<150> US 62/320,231
<151> 2016-04-08
<150> US 62/296,522
<151> 2016-02-17
<150> US 62/285,349
<151> 2015-10-22
<150> US 62/181,675
<151> 2015-06-18
<160> 2233
<170> PatentIn version 3.5
<210> 1
<211> 7
<212> PRT
<213> Simian virus 40
<400> 1
Pro Lys Lys Lys Arg Lys Val
1 5
<210> 2
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Nucleoplasmin bipartite NLS
sequence"
<400> 2
Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys Lys Lys
1 5 10 15
<210> 3
<211> 9
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(9)
<223> /note="Description of Unknown: C-myc NLS sequence"
<400> 3
Pro Ala Ala Lys Arg Val Lys Leu Asp
1 5
<210> 4
<211> 11
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(11)
<223> /note="Description of Unknown: C-myc NLS sequence"
<400> 4
Arg Gln Arg Arg Asn Glu Leu Lys Arg Ser Pro
1 5 10
<210> 5
<211> 38
<212> PRT
<213> Homo sapiens
<400> 5
Asn Gln Ser Ser Asn Phe Gly Pro Met Lys Gly Gly Asn Phe Gly Gly
1 5 10 15
Arg Ser Ser Gly Pro Tyr Gly Gly Gly Gly Gln Tyr Phe Ala Lys Pro
20 25 30
Arg Asn Gln Gly Gly Tyr
35
<210> 6
<211> 42
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(42)
<223> /note="Description of Unknown: IBB domain from importin-alpha
sequence"
<400> 6
Arg Met Arg Ile Glx Phe Lys Asn Lys Gly Lys Asp Thr Ala Glu Leu
1 5 10 15
Arg Arg Arg Arg Val Glu Val Ser Val Glu Leu Arg Lys Ala Lys Lys
20 25 30
Asp Glu Gln Ile Leu Lys Arg Arg Asn Val
35 40
<210> 7
<211> 8
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(8)
<223> /note="Description of Unknown: Myoma T protein sequence"
<400> 7
Val Ser Arg Lys Arg Pro Arg Pro
1 5
<210> 8
<211> 8
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(8)
<223> /note="Description of Unknown: Myoma T protein sequence"
<400> 8
Pro Pro Lys Lys Ala Arg Glu Asp
1 5
<210> 9
<211> 8
<212> PRT
<213> Homo sapiens
<400> 9
Pro Gln Pro Lys Lys Lys Pro Leu
1 5
<210> 10
<211> 12
<212> PRT
<213> Mus musculus
<400> 10
Ser Ala Leu Ile Lys Lys Lys Lys Lys Met Ala Pro
1 5 10
<210> 11
<211> 5
<212> PRT
<213> Influenza virus
<400> 11
Asp Arg Leu Arg Arg
1 5
<210> 12
<211> 7
<212> PRT
<213> Influenza virus
<400> 12
Pro Lys Gln Lys Lys Arg Lys
1 5
<210> 13
<211> 10
<212> PRT
<213> Hepatitis delta virus
<400> 13
Arg Lys Leu Lys Lys Lys Ile Lys Lys Leu
1 5 10
<210> 14
<211> 10
<212> PRT
<213> Mus musculus
<400> 14
Arg Glu Lys Lys Lys Phe Leu Lys Arg Arg
1 5 10
<210> 15
<211> 20
<212> PRT
<213> Homo sapiens
<400> 15
Lys Arg Lys Gly Asp Glu Val Asp Gly Val Asp Glu Val Ala Lys Lys
1 5 10 15
Lys Ser Lys Lys
20
<210> 16
<211> 17
<212> PRT
<213> Homo sapiens
<400> 16
Arg Lys Cys Leu Gln Ala Gly Met Asn Leu Glu Ala Arg Lys Thr Lys
1 5 10 15
Lys
<210> 17
<211> 4
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(4)
<223> /note="Description of Unknown: 'KDEL' family motif peptide"
<400> 17
Lys Asp Glu Leu
1
<210> 18
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 18
Gly Gly Gly Ser
1
<210> 19
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 19
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 20
<211> 30
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 20
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
20 25 30
<210> 21
<211> 45
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 21
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
20 25 30
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
35 40 45
<210> 22
<211> 60
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 22
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
20 25 30
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
35 40 45
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
50 55 60
<210> 23
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> MOD_RES
<222> (2)..(2)
<223> Aminohexanoyl
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Aminohexanoyl
<220>
<221> MOD_RES
<222> (8)..(8)
<223> Aminohexanoyl
<220>
<221> MOD_RES
<222> (11)..(11)
<223> Aminohexanoyl
<400> 23
Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg Arg Xaa Arg
1 5 10
<210> 24
<211> 20
<212> DNA
<213> Homo sapiens
<400> 24
gagtccgagc agaagaagaa 20
<210> 25
<211> 20
<212> DNA
<213> Homo sapiens
<400> 25
gagtcctagc aggagaagaa 20
<210> 26
<211> 20
<212> DNA
<213> Homo sapiens
<400> 26
gagtctaagc agaagaagaa 20
<210> 27
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 27
gccgcagcga augccguuuc acgaaucguc aggcgg 36
<210> 28
<211> 75
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 28
gcuggagacg uuuuuugaaa cggcgagugc ugcggauagc gaguuucucu uggggaggcg 60
cucgcggcca cuuuu 75
<210> 29
<211> 1388
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(1388)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 29
Met Ser Leu Asn Arg Ile Tyr Gln Gly Arg Val Ala Ala Val Glu Thr
1 5 10 15
Gly Thr Ala Leu Ala Lys Gly Asn Val Glu Trp Met Pro Ala Ala Gly
20 25 30
Gly Asp Glu Val Leu Trp Gln His His Glu Leu Phe Gln Ala Ala Ile
35 40 45
Asn Tyr Tyr Leu Val Ala Leu Leu Ala Leu Ala Asp Lys Asn Asn Pro
50 55 60
Val Leu Gly Pro Leu Ile Ser Gln Met Asp Asn Pro Gln Ser Pro Tyr
65 70 75 80
His Val Trp Gly Ser Phe Arg Arg Gln Gly Arg Gln Arg Thr Gly Leu
85 90 95
Ser Gln Ala Val Ala Pro Tyr Ile Thr Pro Gly Asn Asn Ala Pro Thr
100 105 110
Leu Asp Glu Val Phe Arg Ser Ile Leu Ala Gly Asn Pro Thr Asp Arg
115 120 125
Ala Thr Leu Asp Ala Ala Leu Met Gln Leu Leu Lys Ala Cys Asp Gly
130 135 140
Ala Gly Ala Ile Gln Gln Glu Gly Arg Ser Tyr Trp Pro Lys Phe Cys
145 150 155 160
Asp Pro Asp Ser Thr Ala Asn Phe Ala Gly Asp Pro Ala Met Leu Arg
165 170 175
Arg Glu Gln His Arg Leu Leu Leu Pro Gln Val Leu His Asp Pro Ala
180 185 190
Ile Thr His Asp Ser Pro Ala Leu Gly Ser Phe Asp Thr Tyr Ser Ile
195 200 205
Ala Thr Pro Asp Thr Arg Thr Pro Gln Leu Thr Gly Pro Lys Ala Arg
210 215 220
Ala Arg Leu Glu Gln Ala Ile Thr Leu Trp Arg Val Arg Leu Pro Glu
225 230 235 240
Ser Ala Ala Asp Phe Asp Arg Leu Ala Ser Ser Leu Lys Lys Ile Pro
245 250 255
Asp Asp Asp Ser Arg Leu Asn Leu Gln Gly Tyr Val Gly Ser Ser Ala
260 265 270
Lys Gly Glu Val Gln Ala Arg Leu Phe Ala Leu Leu Leu Phe Arg His
275 280 285
Leu Glu Arg Ser Ser Phe Thr Leu Gly Leu Leu Arg Ser Ala Thr Pro
290 295 300
Pro Pro Lys Asn Ala Glu Thr Pro Pro Pro Ala Gly Val Pro Leu Pro
305 310 315 320
Ala Ala Ser Ala Ala Asp Pro Val Arg Ile Ala Arg Gly Lys Arg Ser
325 330 335
Phe Val Phe Arg Ala Phe Thr Ser Leu Pro Cys Trp His Gly Gly Asp
340 345 350
Asn Ile His Pro Thr Trp Lys Ser Phe Asp Ile Ala Ala Phe Lys Tyr
355 360 365
Ala Leu Thr Val Ile Asn Gln Ile Glu Glu Lys Thr Lys Glu Arg Gln
370 375 380
Lys Glu Cys Ala Glu Leu Glu Thr Asp Phe Asp Tyr Met His Gly Arg
385 390 395 400
Leu Ala Lys Ile Pro Val Lys Tyr Thr Thr Gly Glu Ala Glu Pro Pro
405 410 415
Pro Ile Leu Ala Asn Asp Leu Arg Ile Pro Leu Leu Arg Glu Leu Leu
420 425 430
Gln Asn Ile Lys Val Asp Thr Ala Leu Thr Asp Gly Glu Ala Val Ser
435 440 445
Tyr Gly Leu Gln Arg Arg Thr Ile Arg Gly Phe Arg Glu Leu Arg Arg
450 455 460
Ile Trp Arg Gly His Ala Pro Ala Gly Thr Val Phe Ser Ser Glu Leu
465 470 475 480
Lys Glu Lys Leu Ala Gly Glu Leu Arg Gln Phe Gln Thr Asp Asn Ser
485 490 495
Thr Thr Ile Gly Ser Val Gln Leu Phe Asn Glu Leu Ile Gln Asn Pro
500 505 510
Lys Tyr Trp Pro Ile Trp Gln Ala Pro Asp Val Glu Thr Ala Arg Gln
515 520 525
Trp Ala Asp Ala Gly Phe Ala Asp Asp Pro Leu Ala Ala Leu Val Gln
530 535 540
Glu Ala Glu Leu Gln Glu Asp Ile Asp Ala Leu Lys Ala Pro Val Lys
545 550 555 560
Leu Thr Pro Ala Asp Pro Glu Tyr Ser Arg Arg Gln Tyr Asp Phe Asn
565 570 575
Ala Val Ser Lys Phe Gly Ala Gly Ser Arg Ser Ala Asn Arg His Glu
580 585 590
Pro Gly Gln Thr Glu Arg Gly His Asn Thr Phe Thr Thr Glu Ile Ala
595 600 605
Ala Arg Asn Ala Ala Asp Gly Asn Arg Trp Arg Ala Thr His Val Arg
610 615 620
Ile His Tyr Ser Ala Pro Arg Leu Leu Arg Asp Gly Leu Arg Arg Pro
625 630 635 640
Asp Thr Asp Gly Asn Glu Ala Leu Glu Ala Val Pro Trp Leu Gln Pro
645 650 655
Met Met Glu Ala Leu Ala Pro Leu Pro Thr Leu Pro Gln Asp Leu Thr
660 665 670
Gly Met Pro Val Phe Leu Met Pro Asp Val Thr Leu Ser Gly Glu Arg
675 680 685
Arg Ile Leu Leu Asn Leu Pro Val Thr Leu Glu Pro Ala Ala Leu Val
690 695 700
Glu Gln Leu Gly Asn Ala Gly Arg Trp Gln Asn Gln Phe Phe Gly Ser
705 710 715 720
Arg Glu Asp Pro Phe Ala Leu Arg Trp Pro Ala Asp Gly Ala Val Lys
725 730 735
Thr Ala Lys Gly Lys Thr His Ile Pro Trp His Gln Asp Arg Asp His
740 745 750
Phe Thr Val Leu Gly Val Asp Leu Gly Thr Arg Asp Ala Gly Ala Leu
755 760 765
Ala Leu Leu Asn Val Thr Ala Gln Lys Pro Ala Lys Pro Val His Arg
770 775 780
Ile Ile Gly Glu Ala Asp Gly Arg Thr Trp Tyr Ala Ser Leu Ala Asp
785 790 795 800
Ala Arg Met Ile Arg Leu Pro Gly Glu Asp Ala Arg Leu Phe Val Arg
805 810 815
Gly Lys Leu Val Gln Glu Pro Tyr Gly Glu Arg Gly Arg Asn Ala Ser
820 825 830
Leu Leu Glu Trp Glu Asp Ala Arg Asn Ile Ile Leu Arg Leu Gly Gln
835 840 845
Asn Pro Asp Glu Leu Leu Gly Ala Asp Pro Arg Arg His Ser Tyr Pro
850 855 860
Glu Ile Asn Asp Lys Leu Leu Val Ala Leu Arg Arg Ala Gln Ala Arg
865 870 875 880
Leu Ala Arg Leu Gln Asn Arg Ser Trp Arg Leu Arg Asp Leu Ala Glu
885 890 895
Ser Asp Lys Ala Leu Asp Glu Ile His Ala Glu Arg Ala Gly Glu Lys
900 905 910
Pro Ser Pro Leu Pro Pro Leu Ala Arg Asp Asp Ala Ile Lys Ser Thr
915 920 925
Asp Glu Ala Leu Leu Ser Gln Arg Asp Ile Ile Arg Arg Ser Phe Val
930 935 940
Gln Ile Ala Asn Leu Ile Leu Pro Leu Arg Gly Arg Arg Trp Glu Trp
945 950 955 960
Arg Pro His Val Glu Val Pro Asp Cys His Ile Leu Ala Gln Ser Asp
965 970 975
Pro Gly Thr Asp Asp Thr Lys Arg Leu Val Ala Gly Gln Arg Gly Ile
980 985 990
Ser His Glu Arg Ile Glu Gln Ile Glu Glu Leu Arg Arg Arg Cys Gln
995 1000 1005
Ser Leu Asn Arg Ala Leu Arg His Lys Pro Gly Glu Arg Pro Val
1010 1015 1020
Leu Gly Arg Pro Ala Lys Gly Glu Glu Ile Ala Asp Pro Cys Pro
1025 1030 1035
Ala Leu Leu Glu Lys Ile Asn Arg Leu Arg Asp Gln Arg Val Asp
1040 1045 1050
Gln Thr Ala His Ala Ile Leu Ala Ala Ala Leu Gly Val Arg Leu
1055 1060 1065
Arg Ala Pro Ser Lys Asp Arg Ala Glu Arg Arg His Arg Asp Ile
1070 1075 1080
His Gly Glu Tyr Glu Arg Phe Arg Ala Pro Ala Asp Phe Val Val
1085 1090 1095
Ile Glu Asn Leu Ser Arg Tyr Leu Ser Ser Gln Asp Arg Ala Arg
1100 1105 1110
Ser Glu Asn Thr Arg Leu Met Gln Trp Cys His Arg Gln Ile Val
1115 1120 1125
Gln Lys Leu Arg Gln Leu Cys Glu Thr Tyr Gly Ile Pro Val Leu
1130 1135 1140
Ala Val Pro Ala Ala Tyr Ser Ser Arg Phe Ser Ser Arg Asp Gly
1145 1150 1155
Ser Ala Gly Phe Arg Ala Val His Leu Thr Pro Asp His Arg His
1160 1165 1170
Arg Met Pro Trp Ser Arg Ile Leu Ala Arg Leu Lys Ala His Glu
1175 1180 1185
Glu Asp Gly Lys Arg Leu Glu Lys Thr Val Leu Asp Glu Ala Arg
1190 1195 1200
Ala Val Arg Gly Leu Phe Asp Arg Leu Asp Arg Phe Asn Ala Gly
1205 1210 1215
His Val Pro Gly Lys Pro Trp Arg Thr Leu Leu Ala Pro Leu Pro
1220 1225 1230
Gly Gly Pro Val Phe Val Pro Leu Gly Asp Ala Thr Pro Met Gln
1235 1240 1245
Ala Asp Leu Asn Ala Ala Ile Asn Ile Ala Leu Arg Gly Ile Ala
1250 1255 1260
Ala Pro Asp Arg His Asp Ile His His Arg Leu Arg Ala Glu Asn
1265 1270 1275
Lys Lys Arg Ile Leu Ser Leu Arg Leu Gly Thr Gln Arg Glu Lys
1280 1285 1290
Ala Arg Trp Pro Gly Gly Ala Pro Ala Val Thr Leu Ser Thr Pro
1295 1300 1305
Asn Asn Gly Ala Ser Pro Glu Asp Ser Asp Ala Leu Pro Glu Arg
1310 1315 1320
Val Ser Asn Leu Phe Val Asp Ile Ala Gly Val Ala Asn Phe Glu
1325 1330 1335
Arg Val Thr Ile Glu Gly Val Ser Gln Lys Phe Ala Thr Gly Arg
1340 1345 1350
Gly Leu Trp Ala Ser Val Lys Gln Arg Ala Trp Asn Arg Val Ala
1355 1360 1365
Arg Leu Asn Glu Thr Val Thr Asp Asn Asn Arg Asn Glu Glu Glu
1370 1375 1380
Asp Asp Ile Pro Met
1385
<210> 30
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 30
guccaagaaa aaagaaauga uacgaggcau uagcac 36
<210> 31
<211> 107
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 31
cuggacgaug ucucuuuuau uucuuuuuuc uuggaucuga guacgagcac ccacauugga 60
cauuucgcau ggugggugcu cguacuauag guaaaacaaa ccuuuuu 107
<210> 32
<211> 1108
<212> PRT
<213> Bacillus thermoamylovorans
<400> 32
Met Ala Thr Arg Ser Phe Ile Leu Lys Ile Glu Pro Asn Glu Glu Val
1 5 10 15
Lys Lys Gly Leu Trp Lys Thr His Glu Val Leu Asn His Gly Ile Ala
20 25 30
Tyr Tyr Met Asn Ile Leu Lys Leu Ile Arg Gln Glu Ala Ile Tyr Glu
35 40 45
His His Glu Gln Asp Pro Lys Asn Pro Lys Lys Val Ser Lys Ala Glu
50 55 60
Ile Gln Ala Glu Leu Trp Asp Phe Val Leu Lys Met Gln Lys Cys Asn
65 70 75 80
Ser Phe Thr His Glu Val Asp Lys Asp Val Val Phe Asn Ile Leu Arg
85 90 95
Glu Leu Tyr Glu Glu Leu Val Pro Ser Ser Val Glu Lys Lys Gly Glu
100 105 110
Ala Asn Gln Leu Ser Asn Lys Phe Leu Tyr Pro Leu Val Asp Pro Asn
115 120 125
Ser Gln Ser Gly Lys Gly Thr Ala Ser Ser Gly Arg Lys Pro Arg Trp
130 135 140
Tyr Asn Leu Lys Ile Ala Gly Asp Pro Ser Trp Glu Glu Glu Lys Lys
145 150 155 160
Lys Trp Glu Glu Asp Lys Lys Lys Asp Pro Leu Ala Lys Ile Leu Gly
165 170 175
Lys Leu Ala Glu Tyr Gly Leu Ile Pro Leu Phe Ile Pro Phe Thr Asp
180 185 190
Ser Asn Glu Pro Ile Val Lys Glu Ile Lys Trp Met Glu Lys Ser Arg
195 200 205
Asn Gln Ser Val Arg Arg Leu Asp Lys Asp Met Phe Ile Gln Ala Leu
210 215 220
Glu Arg Phe Leu Ser Trp Glu Ser Trp Asn Leu Lys Val Lys Glu Glu
225 230 235 240
Tyr Glu Lys Val Glu Lys Glu His Lys Thr Leu Glu Glu Arg Ile Lys
245 250 255
Glu Asp Ile Gln Ala Phe Lys Ser Leu Glu Gln Tyr Glu Lys Glu Arg
260 265 270
Gln Glu Gln Leu Leu Arg Asp Thr Leu Asn Thr Asn Glu Tyr Arg Leu
275 280 285
Ser Lys Arg Gly Leu Arg Gly Trp Arg Glu Ile Ile Gln Lys Trp Leu
290 295 300
Lys Met Asp Glu Asn Glu Pro Ser Glu Lys Tyr Leu Glu Val Phe Lys
305 310 315 320
Asp Tyr Gln Arg Lys His Pro Arg Glu Ala Gly Asp Tyr Ser Val Tyr
325 330 335
Glu Phe Leu Ser Lys Lys Glu Asn His Phe Ile Trp Arg Asn His Pro
340 345 350
Glu Tyr Pro Tyr Leu Tyr Ala Thr Phe Cys Glu Ile Asp Lys Lys Lys
355 360 365
Lys Asp Ala Lys Gln Gln Ala Thr Phe Thr Leu Ala Asp Pro Ile Asn
370 375 380
His Pro Leu Trp Val Arg Phe Glu Glu Arg Ser Gly Ser Asn Leu Asn
385 390 395 400
Lys Tyr Arg Ile Leu Thr Glu Gln Leu His Thr Glu Lys Leu Lys Lys
405 410 415
Lys Leu Thr Val Gln Leu Asp Arg Leu Ile Tyr Pro Thr Glu Ser Gly
420 425 430
Gly Trp Glu Glu Lys Gly Lys Val Asp Ile Val Leu Leu Pro Ser Arg
435 440 445
Gln Phe Tyr Asn Gln Ile Phe Leu Asp Ile Glu Glu Lys Gly Lys His
450 455 460
Ala Phe Thr Tyr Lys Asp Glu Ser Ile Lys Phe Pro Leu Lys Gly Thr
465 470 475 480
Leu Gly Gly Ala Arg Val Gln Phe Asp Arg Asp His Leu Arg Arg Tyr
485 490 495
Pro His Lys Val Glu Ser Gly Asn Val Gly Arg Ile Tyr Phe Asn Met
500 505 510
Thr Val Asn Ile Glu Pro Thr Glu Ser Pro Val Ser Lys Ser Leu Lys
515 520 525
Ile His Arg Asp Asp Phe Pro Lys Phe Val Asn Phe Lys Pro Lys Glu
530 535 540
Leu Thr Glu Trp Ile Lys Asp Ser Lys Gly Lys Lys Leu Lys Ser Gly
545 550 555 560
Ile Glu Ser Leu Glu Ile Gly Leu Arg Val Met Ser Ile Asp Leu Gly
565 570 575
Gln Arg Gln Ala Ala Ala Ala Ser Ile Phe Glu Val Val Asp Gln Lys
580 585 590
Pro Asp Ile Glu Gly Lys Leu Phe Phe Pro Ile Lys Gly Thr Glu Leu
595 600 605
Tyr Ala Val His Arg Ala Ser Phe Asn Ile Lys Leu Pro Gly Glu Thr
610 615 620
Leu Val Lys Ser Arg Glu Val Leu Arg Lys Ala Arg Glu Asp Asn Leu
625 630 635 640
Lys Leu Met Asn Gln Lys Leu Asn Phe Leu Arg Asn Val Leu His Phe
645 650 655
Gln Gln Phe Glu Asp Ile Thr Glu Arg Glu Lys Arg Val Thr Lys Trp
660 665 670
Ile Ser Arg Gln Glu Asn Ser Asp Val Pro Leu Val Tyr Gln Asp Glu
675 680 685
Leu Ile Gln Ile Arg Glu Leu Met Tyr Lys Pro Tyr Lys Asp Trp Val
690 695 700
Ala Phe Leu Lys Gln Leu His Lys Arg Leu Glu Val Glu Ile Gly Lys
705 710 715 720
Glu Val Lys His Trp Arg Lys Ser Leu Ser Asp Gly Arg Lys Gly Leu
725 730 735
Tyr Gly Ile Ser Leu Lys Asn Ile Asp Glu Ile Asp Arg Thr Arg Lys
740 745 750
Phe Leu Leu Arg Trp Ser Leu Arg Pro Thr Glu Pro Gly Glu Val Arg
755 760 765
Arg Leu Glu Pro Gly Gln Arg Phe Ala Ile Asp Gln Leu Asn His Leu
770 775 780
Asn Ala Leu Lys Glu Asp Arg Leu Lys Lys Met Ala Asn Thr Ile Ile
785 790 795 800
Met His Ala Leu Gly Tyr Cys Tyr Asp Val Arg Lys Lys Lys Trp Gln
805 810 815
Ala Lys Asn Pro Ala Cys Gln Ile Ile Leu Phe Glu Asp Leu Ser Asn
820 825 830
Tyr Asn Pro Tyr Glu Glu Arg Ser Arg Phe Glu Asn Ser Lys Leu Met
835 840 845
Lys Trp Ser Arg Arg Glu Ile Pro Arg Gln Val Ala Leu Gln Gly Glu
850 855 860
Ile Tyr Gly Leu Gln Val Gly Glu Val Gly Ala Gln Phe Ser Ser Arg
865 870 875 880
Phe His Ala Lys Thr Gly Ser Pro Gly Ile Arg Cys Ser Val Val Thr
885 890 895
Lys Glu Lys Leu Gln Asp Asn Arg Phe Phe Lys Asn Leu Gln Arg Glu
900 905 910
Gly Arg Leu Thr Leu Asp Lys Ile Ala Val Leu Lys Glu Gly Asp Leu
915 920 925
Tyr Pro Asp Lys Gly Gly Glu Lys Phe Ile Ser Leu Ser Lys Asp Arg
930 935 940
Lys Leu Val Thr Thr His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln
945 950 955 960
Lys Arg Phe Trp Thr Arg Thr His Gly Phe Tyr Lys Val Tyr Cys Lys
965 970 975
Ala Tyr Gln Val Asp Gly Gln Thr Val Tyr Ile Pro Glu Ser Lys Asp
980 985 990
Gln Lys Gln Lys Ile Ile Glu Glu Phe Gly Glu Gly Tyr Phe Ile Leu
995 1000 1005
Lys Asp Gly Val Tyr Glu Trp Gly Asn Ala Gly Lys Leu Lys Ile
1010 1015 1020
Lys Lys Gly Ser Ser Lys Gln Ser Ser Ser Glu Leu Val Asp Ser
1025 1030 1035
Asp Ile Leu Lys Asp Ser Phe Asp Leu Ala Ser Glu Leu Lys Gly
1040 1045 1050
Glu Lys Leu Met Leu Tyr Arg Asp Pro Ser Gly Asn Val Phe Pro
1055 1060 1065
Ser Asp Lys Trp Met Ala Ala Gly Val Phe Phe Gly Lys Leu Glu
1070 1075 1080
Arg Ile Leu Ile Ser Lys Leu Thr Asn Gln Tyr Ser Ile Ser Thr
1085 1090 1095
Ile Glu Asp Asp Ser Ser Lys Gln Ser Met
1100 1105
<210> 33
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 33
guucgaaagc uuaguggaaa gcuucguggu uagcac 36
<210> 34
<211> 69
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 34
cacggauaau cacgacuuuc cacuaagcuu ucgaauuuua ugaugcgagc auccucucag 60
gucaaaaaa 69
<210> 35
<211> 1108
<212> PRT
<213> Bacillus sp.
<400> 35
Met Ala Ile Arg Ser Ile Lys Leu Lys Leu Lys Thr His Thr Gly Pro
1 5 10 15
Glu Ala Gln Asn Leu Arg Lys Gly Ile Trp Arg Thr His Arg Leu Leu
20 25 30
Asn Glu Gly Val Ala Tyr Tyr Met Lys Met Leu Leu Leu Phe Arg Gln
35 40 45
Glu Ser Thr Gly Glu Arg Pro Lys Glu Glu Leu Gln Glu Glu Leu Ile
50 55 60
Cys His Ile Arg Glu Gln Gln Gln Arg Asn Gln Ala Asp Lys Asn Thr
65 70 75 80
Gln Ala Leu Pro Leu Asp Lys Ala Leu Glu Ala Leu Arg Gln Leu Tyr
85 90 95
Glu Leu Leu Val Pro Ser Ser Val Gly Gln Ser Gly Asp Ala Gln Ile
100 105 110
Ile Ser Arg Lys Phe Leu Ser Pro Leu Val Asp Pro Asn Ser Glu Gly
115 120 125
Gly Lys Gly Thr Ser Lys Ala Gly Ala Lys Pro Thr Trp Gln Lys Lys
130 135 140
Lys Glu Ala Asn Asp Pro Thr Trp Glu Gln Asp Tyr Glu Lys Trp Lys
145 150 155 160
Lys Arg Arg Glu Glu Asp Pro Thr Ala Ser Val Ile Thr Thr Leu Glu
165 170 175
Glu Tyr Gly Ile Arg Pro Ile Phe Pro Leu Tyr Thr Asn Thr Val Thr
180 185 190
Asp Ile Ala Trp Leu Pro Leu Gln Ser Asn Gln Phe Val Arg Thr Trp
195 200 205
Asp Arg Asp Met Leu Gln Gln Ala Ile Glu Arg Leu Leu Ser Trp Glu
210 215 220
Ser Trp Asn Lys Arg Val Gln Glu Glu Tyr Ala Lys Leu Lys Glu Lys
225 230 235 240
Met Ala Gln Leu Asn Glu Gln Leu Glu Gly Gly Gln Glu Trp Ile Ser
245 250 255
Leu Leu Glu Gln Tyr Glu Glu Asn Arg Glu Arg Glu Leu Arg Glu Asn
260 265 270
Met Thr Ala Ala Asn Asp Lys Tyr Arg Ile Thr Lys Arg Gln Met Lys
275 280 285
Gly Trp Asn Glu Leu Tyr Glu Leu Trp Ser Thr Phe Pro Ala Ser Ala
290 295 300
Ser His Glu Gln Tyr Lys Glu Ala Leu Lys Arg Val Gln Gln Arg Leu
305 310 315 320
Arg Gly Arg Phe Gly Asp Ala His Phe Phe Gln Tyr Leu Met Glu Glu
325 330 335
Lys Asn Arg Leu Ile Trp Lys Gly Asn Pro Gln Arg Ile His Tyr Phe
340 345 350
Val Ala Arg Asn Glu Leu Thr Lys Arg Leu Glu Glu Ala Lys Gln Ser
355 360 365
Ala Thr Met Thr Leu Pro Asn Ala Arg Lys His Pro Leu Trp Val Arg
370 375 380
Phe Asp Ala Arg Gly Gly Asn Leu Gln Asp Tyr Tyr Leu Thr Ala Glu
385 390 395 400
Ala Asp Lys Pro Arg Ser Arg Arg Phe Val Thr Phe Ser Gln Leu Ile
405 410 415
Trp Pro Ser Glu Ser Gly Trp Met Glu Lys Lys Asp Val Glu Val Glu
420 425 430
Leu Ala Leu Ser Arg Gln Phe Tyr Gln Gln Val Lys Leu Leu Lys Asn
435 440 445
Asp Lys Gly Lys Gln Lys Ile Glu Phe Lys Asp Lys Gly Ser Gly Ser
450 455 460
Thr Phe Asn Gly His Leu Gly Gly Ala Lys Leu Gln Leu Glu Arg Gly
465 470 475 480
Asp Leu Glu Lys Glu Glu Lys Asn Phe Glu Asp Gly Glu Ile Gly Ser
485 490 495
Val Tyr Leu Asn Val Val Ile Asp Phe Glu Pro Leu Gln Glu Val Lys
500 505 510
Asn Gly Arg Val Gln Ala Pro Tyr Gly Gln Val Leu Gln Leu Ile Arg
515 520 525
Arg Pro Asn Glu Phe Pro Lys Val Thr Thr Tyr Lys Ser Glu Gln Leu
530 535 540
Val Glu Trp Ile Lys Ala Ser Pro Gln His Ser Ala Gly Val Glu Ser
545 550 555 560
Leu Ala Ser Gly Phe Arg Val Met Ser Ile Asp Leu Gly Leu Arg Ala
565 570 575
Ala Ala Ala Thr Ser Ile Phe Ser Val Glu Glu Ser Ser Asp Lys Asn
580 585 590
Ala Ala Asp Phe Ser Tyr Trp Ile Glu Gly Thr Pro Leu Val Ala Val
595 600 605
His Gln Arg Ser Tyr Met Leu Arg Leu Pro Gly Glu Gln Val Glu Lys
610 615 620
Gln Val Met Glu Lys Arg Asp Glu Arg Phe Gln Leu His Gln Arg Val
625 630 635 640
Lys Phe Gln Ile Arg Val Leu Ala Gln Ile Met Arg Met Ala Asn Lys
645 650 655
Gln Tyr Gly Asp Arg Trp Asp Glu Leu Asp Ser Leu Lys Gln Ala Val
660 665 670
Glu Gln Lys Lys Ser Pro Leu Asp Gln Thr Asp Arg Thr Phe Trp Glu
675 680 685
Gly Ile Val Cys Asp Leu Thr Lys Val Leu Pro Arg Asn Glu Ala Asp
690 695 700
Trp Glu Gln Ala Val Val Gln Ile His Arg Lys Ala Glu Glu Tyr Val
705 710 715 720
Gly Lys Ala Val Gln Ala Trp Arg Lys Arg Phe Ala Ala Asp Glu Arg
725 730 735
Lys Gly Ile Ala Gly Leu Ser Met Trp Asn Ile Glu Glu Leu Glu Gly
740 745 750
Leu Arg Lys Leu Leu Ile Ser Trp Ser Arg Arg Thr Arg Asn Pro Gln
755 760 765
Glu Val Asn Arg Phe Glu Arg Gly His Thr Ser His Gln Arg Leu Leu
770 775 780
Thr His Ile Gln Asn Val Lys Glu Asp Arg Leu Lys Gln Leu Ser His
785 790 795 800
Ala Ile Val Met Thr Ala Leu Gly Tyr Val Tyr Asp Glu Arg Lys Gln
805 810 815
Glu Trp Cys Ala Glu Tyr Pro Ala Cys Gln Val Ile Leu Phe Glu Asn
820 825 830
Leu Ser Gln Tyr Arg Ser Asn Leu Asp Arg Ser Thr Lys Glu Asn Ser
835 840 845
Thr Leu Met Lys Trp Ala His Arg Ser Ile Pro Lys Tyr Val His Met
850 855 860
Gln Ala Glu Pro Tyr Gly Ile Gln Ile Gly Asp Val Arg Ala Glu Tyr
865 870 875 880
Ser Ser Arg Phe Tyr Ala Lys Thr Gly Thr Pro Gly Ile Arg Cys Lys
885 890 895
Lys Val Arg Gly Gln Asp Leu Gln Gly Arg Arg Phe Glu Asn Leu Gln
900 905 910
Lys Arg Leu Val Asn Glu Gln Phe Leu Thr Glu Glu Gln Val Lys Gln
915 920 925
Leu Arg Pro Gly Asp Ile Val Pro Asp Asp Ser Gly Glu Leu Phe Met
930 935 940
Thr Leu Thr Asp Gly Ser Gly Ser Lys Glu Val Val Phe Leu Gln Ala
945 950 955 960
Asp Ile Asn Ala Ala His Asn Leu Gln Lys Arg Phe Trp Gln Arg Tyr
965 970 975
Asn Glu Leu Phe Lys Val Ser Cys Arg Val Ile Val Arg Asp Glu Glu
980 985 990
Glu Tyr Leu Val Pro Lys Thr Lys Ser Val Gln Ala Lys Leu Gly Lys
995 1000 1005
Gly Leu Phe Val Lys Lys Ser Asp Thr Ala Trp Lys Asp Val Tyr
1010 1015 1020
Val Trp Asp Ser Gln Ala Lys Leu Lys Gly Lys Thr Thr Phe Thr
1025 1030 1035
Glu Glu Ser Glu Ser Pro Glu Gln Leu Glu Asp Phe Gln Glu Ile
1040 1045 1050
Ile Glu Glu Ala Glu Glu Ala Lys Gly Thr Tyr Arg Thr Leu Phe
1055 1060 1065
Arg Asp Pro Ser Gly Val Phe Phe Pro Glu Ser Val Trp Tyr Pro
1070 1075 1080
Gln Lys Asp Phe Trp Gly Glu Val Lys Arg Lys Leu Tyr Gly Lys
1085 1090 1095
Leu Arg Glu Arg Phe Leu Thr Lys Ala Arg
1100 1105
<210> 36
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 36
guuuugagaa uagcccgaca uagagggcaa uagac 35
<210> 37
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 37
guuaugaaaa cagcccgaca uagagggcaa uagaca 36
<210> 38
<211> 1334
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(1334)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 38
Met Lys Ile Ser Lys Val Asp His Thr Arg Met Ala Val Ala Lys Gly
1 5 10 15
Asn Gln His Arg Arg Asp Glu Ile Ser Gly Ile Leu Tyr Lys Asp Pro
20 25 30
Thr Lys Thr Gly Ser Ile Asp Phe Asp Glu Arg Phe Lys Lys Leu Asn
35 40 45
Cys Ser Ala Lys Ile Leu Tyr His Val Phe Asn Gly Ile Ala Glu Gly
50 55 60
Ser Asn Lys Tyr Lys Asn Ile Val Asp Lys Val Asn Asn Asn Leu Asp
65 70 75 80
Arg Val Leu Phe Thr Gly Lys Ser Tyr Asp Arg Lys Ser Ile Ile Asp
85 90 95
Ile Asp Thr Val Leu Arg Asn Val Glu Lys Ile Asn Ala Phe Asp Arg
100 105 110
Ile Ser Thr Glu Glu Arg Glu Gln Ile Ile Asp Asp Leu Leu Glu Ile
115 120 125
Gln Leu Arg Lys Gly Leu Arg Lys Gly Lys Ala Gly Leu Arg Glu Val
130 135 140
Leu Leu Ile Gly Ala Gly Val Ile Val Arg Thr Asp Lys Lys Gln Glu
145 150 155 160
Ile Ala Asp Phe Leu Glu Ile Leu Asp Glu Asp Phe Asn Lys Thr Asn
165 170 175
Gln Ala Lys Asn Ile Lys Leu Ser Ile Glu Asn Gln Gly Leu Val Val
180 185 190
Ser Pro Val Ser Arg Gly Glu Glu Arg Ile Phe Asp Val Ser Gly Ala
195 200 205
Gln Lys Gly Lys Ser Ser Lys Lys Ala Gln Glu Lys Glu Ala Leu Ser
210 215 220
Ala Phe Leu Leu Asp Tyr Ala Asp Leu Asp Lys Asn Val Arg Phe Glu
225 230 235 240
Tyr Leu Arg Lys Ile Arg Arg Leu Ile Asn Leu Tyr Phe Tyr Val Lys
245 250 255
Asn Asp Asp Val Met Ser Leu Thr Glu Ile Pro Ala Glu Val Asn Leu
260 265 270
Glu Lys Asp Phe Asp Ile Trp Arg Asp His Glu Gln Arg Lys Glu Glu
275 280 285
Asn Gly Asp Phe Val Gly Cys Pro Asp Ile Leu Leu Ala Asp Arg Asp
290 295 300
Val Lys Lys Ser Asn Ser Lys Gln Val Lys Ile Ala Glu Arg Gln Leu
305 310 315 320
Arg Glu Ser Ile Arg Glu Lys Asn Ile Lys Arg Tyr Arg Phe Ser Ile
325 330 335
Lys Thr Ile Glu Lys Asp Asp Gly Thr Tyr Phe Phe Ala Asn Lys Gln
340 345 350
Ile Ser Val Phe Trp Ile His Arg Ile Glu Asn Ala Val Glu Arg Ile
355 360 365
Leu Gly Ser Ile Asn Asp Lys Lys Leu Tyr Arg Leu Arg Leu Gly Tyr
370 375 380
Leu Gly Glu Lys Val Trp Lys Asp Ile Leu Asn Phe Leu Ser Ile Lys
385 390 395 400
Tyr Ile Ala Val Gly Lys Ala Val Phe Asn Phe Ala Met Asp Asp Leu
405 410 415
Gln Glu Lys Asp Arg Asp Ile Glu Pro Gly Lys Ile Ser Glu Asn Ala
420 425 430
Val Asn Gly Leu Thr Ser Phe Asp Tyr Glu Gln Ile Lys Ala Asp Glu
435 440 445
Met Leu Gln Arg Glu Val Ala Val Asn Val Ala Phe Ala Ala Asn Asn
450 455 460
Leu Ala Arg Val Thr Val Asp Ile Pro Gln Asn Gly Glu Lys Glu Asp
465 470 475 480
Ile Leu Leu Trp Asn Lys Ser Asp Ile Lys Lys Tyr Lys Lys Asn Ser
485 490 495
Lys Lys Gly Ile Leu Lys Ser Ile Leu Gln Phe Phe Gly Gly Ala Ser
500 505 510
Thr Trp Asn Met Lys Met Phe Glu Ile Ala Tyr His Asp Gln Pro Gly
515 520 525
Asp Tyr Glu Glu Asn Tyr Leu Tyr Asp Ile Ile Gln Ile Ile Tyr Ser
530 535 540
Leu Arg Asn Lys Ser Phe His Phe Lys Thr Tyr Asp His Gly Asp Lys
545 550 555 560
Asn Trp Asn Arg Glu Leu Ile Gly Lys Met Ile Glu His Asp Ala Glu
565 570 575
Arg Val Ile Ser Val Glu Arg Glu Lys Phe His Ser Asn Asn Leu Pro
580 585 590
Met Phe Tyr Lys Asp Ala Asp Leu Lys Lys Ile Leu Asp Leu Leu Tyr
595 600 605
Ser Asp Tyr Ala Gly Arg Ala Ser Gln Val Pro Ala Phe Asn Thr Val
610 615 620
Leu Val Arg Lys Asn Phe Pro Glu Phe Leu Arg Lys Asp Met Gly Tyr
625 630 635 640
Lys Val His Phe Asn Asn Pro Glu Val Glu Asn Gln Trp His Ser Ala
645 650 655
Val Tyr Tyr Leu Tyr Lys Glu Ile Tyr Tyr Asn Leu Phe Leu Arg Asp
660 665 670
Lys Glu Val Lys Asn Leu Phe Tyr Thr Ser Leu Lys Asn Ile Arg Ser
675 680 685
Glu Val Ser Asp Lys Lys Gln Lys Leu Ala Ser Asp Asp Phe Ala Ser
690 695 700
Arg Cys Glu Glu Ile Glu Asp Arg Ser Leu Pro Glu Ile Cys Gln Ile
705 710 715 720
Ile Met Thr Glu Tyr Asn Ala Gln Asn Phe Gly Asn Arg Lys Val Lys
725 730 735
Ser Gln Arg Val Ile Glu Lys Asn Lys Asp Ile Phe Arg His Tyr Lys
740 745 750
Met Leu Leu Ile Lys Thr Leu Ala Gly Ala Phe Ser Leu Tyr Leu Lys
755 760 765
Gln Glu Arg Phe Ala Phe Ile Gly Lys Ala Thr Pro Ile Pro Tyr Glu
770 775 780
Thr Thr Asp Val Lys Asn Phe Leu Pro Glu Trp Lys Ser Gly Met Tyr
785 790 795 800
Ala Ser Phe Val Glu Glu Ile Lys Asn Asn Leu Asp Leu Gln Glu Trp
805 810 815
Tyr Ile Val Gly Arg Phe Leu Asn Gly Arg Met Leu Asn Gln Leu Ala
820 825 830
Gly Ser Leu Arg Ser Tyr Ile Gln Tyr Ala Glu Asp Ile Glu Arg Arg
835 840 845
Ala Ala Glu Asn Arg Asn Lys Leu Phe Ser Lys Pro Asp Glu Lys Ile
850 855 860
Glu Ala Cys Lys Lys Ala Val Arg Val Leu Asp Leu Cys Ile Lys Ile
865 870 875 880
Ser Thr Arg Ile Ser Ala Glu Phe Thr Asp Tyr Phe Asp Ser Glu Asp
885 890 895
Asp Tyr Ala Asp Tyr Leu Glu Lys Tyr Leu Lys Tyr Gln Asp Asp Ala
900 905 910
Ile Lys Glu Leu Ser Gly Ser Ser Tyr Ala Ala Leu Asp His Phe Cys
915 920 925
Asn Lys Asp Asp Leu Lys Phe Asp Ile Tyr Val Asn Ala Gly Gln Lys
930 935 940
Pro Ile Leu Gln Arg Asn Ile Val Met Ala Lys Leu Phe Gly Pro Asp
945 950 955 960
Asn Ile Leu Ser Glu Val Met Glu Lys Val Thr Glu Ser Ala Ile Arg
965 970 975
Glu Tyr Tyr Asp Tyr Leu Lys Lys Val Ser Gly Tyr Arg Val Arg Gly
980 985 990
Lys Cys Ser Thr Glu Lys Glu Gln Glu Asp Leu Leu Lys Phe Gln Arg
995 1000 1005
Leu Lys Asn Ala Val Glu Phe Arg Asp Val Thr Glu Tyr Ala Glu
1010 1015 1020
Val Ile Asn Glu Leu Leu Gly Gln Leu Ile Ser Trp Ser Tyr Leu
1025 1030 1035
Arg Glu Arg Asp Leu Leu Tyr Phe Gln Leu Gly Phe His Tyr Met
1040 1045 1050
Cys Leu Lys Asn Lys Ser Phe Lys Pro Ala Glu Tyr Val Asp Ile
1055 1060 1065
Arg Arg Asn Asn Gly Thr Ile Ile His Asn Ala Ile Leu Tyr Gln
1070 1075 1080
Ile Val Ser Met Tyr Ile Asn Gly Leu Asp Phe Tyr Ser Cys Asp
1085 1090 1095
Lys Glu Gly Lys Thr Leu Lys Pro Ile Glu Thr Gly Lys Gly Val
1100 1105 1110
Gly Ser Lys Ile Gly Gln Phe Ile Lys Tyr Ser Gln Tyr Leu Tyr
1115 1120 1125
Asn Asp Pro Ser Tyr Lys Leu Glu Ile Tyr Asn Ala Gly Leu Glu
1130 1135 1140
Val Phe Glu Asn Ile Asp Glu His Asp Asn Ile Thr Asp Leu Arg
1145 1150 1155
Lys Tyr Val Asp His Phe Lys Tyr Tyr Ala Tyr Gly Asn Lys Met
1160 1165 1170
Ser Leu Leu Asp Leu Tyr Ser Glu Phe Phe Asp Arg Phe Phe Thr
1175 1180 1185
Tyr Asp Met Lys Tyr Gln Lys Asn Val Val Asn Val Leu Glu Asn
1190 1195 1200
Ile Leu Leu Arg His Phe Val Ile Phe Tyr Pro Lys Phe Gly Ser
1205 1210 1215
Gly Lys Lys Asp Val Gly Ile Arg Asp Cys Lys Lys Glu Arg Ala
1220 1225 1230
Gln Ile Glu Ile Ser Glu Gln Ser Leu Thr Ser Glu Asp Phe Met
1235 1240 1245
Phe Lys Leu Asp Asp Lys Ala Gly Glu Glu Ala Lys Lys Phe Pro
1250 1255 1260
Ala Arg Asp Glu Arg Tyr Leu Gln Thr Ile Ala Lys Leu Leu Tyr
1265 1270 1275
Tyr Pro Asn Glu Ile Glu Asp Met Asn Arg Phe Met Lys Lys Gly
1280 1285 1290
Glu Thr Ile Asn Lys Lys Val Gln Phe Asn Arg Lys Lys Lys Ile
1295 1300 1305
Thr Arg Lys Gln Lys Asn Asn Ser Ser Asn Glu Val Leu Ser Ser
1310 1315 1320
Thr Met Gly Tyr Leu Phe Lys Asn Ile Lys Leu
1325 1330
<210> 39
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 39
guuuuagucc ucuuucauau agagguaguc ucuuac 36
<210> 40
<211> 99
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 40
augaaaagag gacuaaaacu gaaagaggac uaaaacacca gauguggaua acuauauuag 60
uggcuauuaa aaauucgucg auauuagaga ggaaacuuu 99
<210> 41
<211> 1120
<212> PRT
<213> Listeria seeligeri
<400> 41
Met Trp Ile Ser Ile Lys Thr Leu Ile His His Leu Gly Val Leu Phe
1 5 10 15
Phe Cys Asp Tyr Met Tyr Asn Arg Arg Glu Lys Lys Ile Ile Glu Val
20 25 30
Lys Thr Met Arg Ile Thr Lys Val Glu Val Asp Arg Lys Lys Val Leu
35 40 45
Ile Ser Arg Asp Lys Asn Gly Gly Lys Leu Val Tyr Glu Asn Glu Met
50 55 60
Gln Asp Asn Thr Glu Gln Ile Met His His Lys Lys Ser Ser Phe Tyr
65 70 75 80
Lys Ser Val Val Asn Lys Thr Ile Cys Arg Pro Glu Gln Lys Gln Met
85 90 95
Lys Lys Leu Val His Gly Leu Leu Gln Glu Asn Ser Gln Glu Lys Ile
100 105 110
Lys Val Ser Asp Val Thr Lys Leu Asn Ile Ser Asn Phe Leu Asn His
115 120 125
Arg Phe Lys Lys Ser Leu Tyr Tyr Phe Pro Glu Asn Ser Pro Asp Lys
130 135 140
Ser Glu Glu Tyr Arg Ile Glu Ile Asn Leu Ser Gln Leu Leu Glu Asp
145 150 155 160
Ser Leu Lys Lys Gln Gln Gly Thr Phe Ile Cys Trp Glu Ser Phe Ser
165 170 175
Lys Asp Met Glu Leu Tyr Ile Asn Trp Ala Glu Asn Tyr Ile Ser Ser
180 185 190
Lys Thr Lys Leu Ile Lys Lys Ser Ile Arg Asn Asn Arg Ile Gln Ser
195 200 205
Thr Glu Ser Arg Ser Gly Gln Leu Met Asp Arg Tyr Met Lys Asp Ile
210 215 220
Leu Asn Lys Asn Lys Pro Phe Asp Ile Gln Ser Val Ser Glu Lys Tyr
225 230 235 240
Gln Leu Glu Lys Leu Thr Ser Ala Leu Lys Ala Thr Phe Lys Glu Ala
245 250 255
Lys Lys Asn Asp Lys Glu Ile Asn Tyr Lys Leu Lys Ser Thr Leu Gln
260 265 270
Asn His Glu Arg Gln Ile Ile Glu Glu Leu Lys Glu Asn Ser Glu Leu
275 280 285
Asn Gln Phe Asn Ile Glu Ile Arg Lys His Leu Glu Thr Tyr Phe Pro
290 295 300
Ile Lys Lys Thr Asn Arg Lys Val Gly Asp Ile Arg Asn Leu Glu Ile
305 310 315 320
Gly Glu Ile Gln Lys Ile Val Asn His Arg Leu Lys Asn Lys Ile Val
325 330 335
Gln Arg Ile Leu Gln Glu Gly Lys Leu Ala Ser Tyr Glu Ile Glu Ser
340 345 350
Thr Val Asn Ser Asn Ser Leu Gln Lys Ile Lys Ile Glu Glu Ala Phe
355 360 365
Ala Leu Lys Phe Ile Asn Ala Cys Leu Phe Ala Ser Asn Asn Leu Arg
370 375 380
Asn Met Val Tyr Pro Val Cys Lys Lys Asp Ile Leu Met Ile Gly Glu
385 390 395 400
Phe Lys Asn Ser Phe Lys Glu Ile Lys His Lys Lys Phe Ile Arg Gln
405 410 415
Trp Ser Gln Phe Phe Ser Gln Glu Ile Thr Val Asp Asp Ile Glu Leu
420 425 430
Ala Ser Trp Gly Leu Arg Gly Ala Ile Ala Pro Ile Arg Asn Glu Ile
435 440 445
Ile His Leu Lys Lys His Ser Trp Lys Lys Phe Phe Asn Asn Pro Thr
450 455 460
Phe Lys Val Lys Lys Ser Lys Ile Ile Asn Gly Lys Thr Lys Asp Val
465 470 475 480
Thr Ser Glu Phe Leu Tyr Lys Glu Thr Leu Phe Lys Asp Tyr Phe Tyr
485 490 495
Ser Glu Leu Asp Ser Val Pro Glu Leu Ile Ile Asn Lys Met Glu Ser
500 505 510
Ser Lys Ile Leu Asp Tyr Tyr Ser Ser Asp Gln Leu Asn Gln Val Phe
515 520 525
Thr Ile Pro Asn Phe Glu Leu Ser Leu Leu Thr Ser Ala Val Pro Phe
530 535 540
Ala Pro Ser Phe Lys Arg Val Tyr Leu Lys Gly Phe Asp Tyr Gln Asn
545 550 555 560
Gln Asp Glu Ala Gln Pro Asp Tyr Asn Leu Lys Leu Asn Ile Tyr Asn
565 570 575
Glu Lys Ala Phe Asn Ser Glu Ala Phe Gln Ala Gln Tyr Ser Leu Phe
580 585 590
Lys Met Val Tyr Tyr Gln Val Phe Leu Pro Gln Phe Thr Thr Asn Asn
595 600 605
Asp Leu Phe Lys Ser Ser Val Asp Phe Ile Leu Thr Leu Asn Lys Glu
610 615 620
Arg Lys Gly Tyr Ala Lys Ala Phe Gln Asp Ile Arg Lys Met Asn Lys
625 630 635 640
Asp Glu Lys Pro Ser Glu Tyr Met Ser Tyr Ile Gln Ser Gln Leu Met
645 650 655
Leu Tyr Gln Lys Lys Gln Glu Glu Lys Glu Lys Ile Asn His Phe Glu
660 665 670
Lys Phe Ile Asn Gln Val Phe Ile Lys Gly Phe Asn Ser Phe Ile Glu
675 680 685
Lys Asn Arg Leu Thr Tyr Ile Cys His Pro Thr Lys Asn Thr Val Pro
690 695 700
Glu Asn Asp Asn Ile Glu Ile Pro Phe His Thr Asp Met Asp Asp Ser
705 710 715 720
Asn Ile Ala Phe Trp Leu Met Cys Lys Leu Leu Asp Ala Lys Gln Leu
725 730 735
Ser Glu Leu Arg Asn Glu Met Ile Lys Phe Ser Cys Ser Leu Gln Ser
740 745 750
Thr Glu Glu Ile Ser Thr Phe Thr Lys Ala Arg Glu Val Ile Gly Leu
755 760 765
Ala Leu Leu Asn Gly Glu Lys Gly Cys Asn Asp Trp Lys Glu Leu Phe
770 775 780
Asp Asp Lys Glu Ala Trp Lys Lys Asn Met Ser Leu Tyr Val Ser Glu
785 790 795 800
Glu Leu Leu Gln Ser Leu Pro Tyr Thr Gln Glu Asp Gly Gln Thr Pro
805 810 815
Val Ile Asn Arg Ser Ile Asp Leu Val Lys Lys Tyr Gly Thr Glu Thr
820 825 830
Ile Leu Glu Lys Leu Phe Ser Ser Ser Asp Asp Tyr Lys Val Ser Ala
835 840 845
Lys Asp Ile Ala Lys Leu His Glu Tyr Asp Val Thr Glu Lys Ile Ala
850 855 860
Gln Gln Glu Ser Leu His Lys Gln Trp Ile Glu Lys Pro Gly Leu Ala
865 870 875 880
Arg Asp Ser Ala Trp Thr Lys Lys Tyr Gln Asn Val Ile Asn Asp Ile
885 890 895
Ser Asn Tyr Gln Trp Ala Lys Thr Lys Val Glu Leu Thr Gln Val Arg
900 905 910
His Leu His Gln Leu Thr Ile Asp Leu Leu Ser Arg Leu Ala Gly Tyr
915 920 925
Met Ser Ile Ala Asp Arg Asp Phe Gln Phe Ser Ser Asn Tyr Ile Leu
930 935 940
Glu Arg Glu Asn Ser Glu Tyr Arg Val Thr Ser Trp Ile Leu Leu Ser
945 950 955 960
Glu Asn Lys Asn Lys Asn Lys Tyr Asn Asp Tyr Glu Leu Tyr Asn Leu
965 970 975
Lys Asn Ala Ser Ile Lys Val Ser Ser Lys Asn Asp Pro Gln Leu Lys
980 985 990
Val Asp Leu Lys Gln Leu Arg Leu Thr Leu Glu Tyr Leu Glu Leu Phe
995 1000 1005
Asp Asn Arg Leu Lys Glu Lys Arg Asn Asn Ile Ser His Phe Asn
1010 1015 1020
Tyr Leu Asn Gly Gln Leu Gly Asn Ser Ile Leu Glu Leu Phe Asp
1025 1030 1035
Asp Ala Arg Asp Val Leu Ser Tyr Asp Arg Lys Leu Lys Asn Ala
1040 1045 1050
Val Ser Lys Ser Leu Lys Glu Ile Leu Ser Ser His Gly Met Glu
1055 1060 1065
Val Thr Phe Lys Pro Leu Tyr Gln Thr Asn His His Leu Lys Ile
1070 1075 1080
Asp Lys Leu Gln Pro Lys Lys Ile His His Leu Gly Glu Lys Ser
1085 1090 1095
Thr Val Ser Ser Asn Gln Val Ser Asn Glu Tyr Cys Gln Leu Val
1100 1105 1110
Arg Thr Leu Leu Thr Met Lys
1115 1120
<210> 42
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 42
guuuuagucc ccuucguuuu ugggguaguc uaaauc 36
<210> 43
<211> 113
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 43
gauuuagagc accccaaaag uaaugaaaau uugcaauuaa auaaggaaua uuaaaaaaau 60
gugauuuuaa aaaaauugaa gaaauuaaau gaaaaauugu ccaaguaaaa aaa 113
<210> 44
<211> 70
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 44
auuuagauua ccccuuuaau uuauuuuacc auauuuuucu cauaaugcaa acuaauauuc 60
caaaauuuuu 70
<210> 45
<211> 1389
<212> PRT
<213> Leptotrichia wadei
<400> 45
Met Gly Asn Leu Phe Gly His Lys Arg Trp Tyr Glu Val Arg Asp Lys
1 5 10 15
Lys Asp Phe Lys Ile Lys Arg Lys Val Lys Val Lys Arg Asn Tyr Asp
20 25 30
Gly Asn Lys Tyr Ile Leu Asn Ile Asn Glu Asn Asn Asn Lys Glu Lys
35 40 45
Ile Asp Asn Asn Lys Phe Ile Arg Lys Tyr Ile Asn Tyr Lys Lys Asn
50 55 60
Asp Asn Ile Leu Lys Glu Phe Thr Arg Lys Phe His Ala Gly Asn Ile
65 70 75 80
Leu Phe Lys Leu Lys Gly Lys Glu Gly Ile Ile Arg Ile Glu Asn Asn
85 90 95
Asp Asp Phe Leu Glu Thr Glu Glu Val Val Leu Tyr Ile Glu Ala Tyr
100 105 110
Gly Lys Ser Glu Lys Leu Lys Ala Leu Gly Ile Thr Lys Lys Lys Ile
115 120 125
Ile Asp Glu Ala Ile Arg Gln Gly Ile Thr Lys Asp Asp Lys Lys Ile
130 135 140
Glu Ile Lys Arg Gln Glu Asn Glu Glu Glu Ile Glu Ile Asp Ile Arg
145 150 155 160
Asp Glu Tyr Thr Asn Lys Thr Leu Asn Asp Cys Ser Ile Ile Leu Arg
165 170 175
Ile Ile Glu Asn Asp Glu Leu Glu Thr Lys Lys Ser Ile Tyr Glu Ile
180 185 190
Phe Lys Asn Ile Asn Met Ser Leu Tyr Lys Ile Ile Glu Lys Ile Ile
195 200 205
Glu Asn Glu Thr Glu Lys Val Phe Glu Asn Arg Tyr Tyr Glu Glu His
210 215 220
Leu Arg Glu Lys Leu Leu Lys Asp Asp Lys Ile Asp Val Ile Leu Thr
225 230 235 240
Asn Phe Met Glu Ile Arg Glu Lys Ile Lys Ser Asn Leu Glu Ile Leu
245 250 255
Gly Phe Val Lys Phe Tyr Leu Asn Val Gly Gly Asp Lys Lys Lys Ser
260 265 270
Lys Asn Lys Lys Met Leu Val Glu Lys Ile Leu Asn Ile Asn Val Asp
275 280 285
Leu Thr Val Glu Asp Ile Ala Asp Phe Val Ile Lys Glu Leu Glu Phe
290 295 300
Trp Asn Ile Thr Lys Arg Ile Glu Lys Val Lys Lys Val Asn Asn Glu
305 310 315 320
Phe Leu Glu Lys Arg Arg Asn Arg Thr Tyr Ile Lys Ser Tyr Val Leu
325 330 335
Leu Asp Lys His Glu Lys Phe Lys Ile Glu Arg Glu Asn Lys Lys Asp
340 345 350
Lys Ile Val Lys Phe Phe Val Glu Asn Ile Lys Asn Asn Ser Ile Lys
355 360 365
Glu Lys Ile Glu Lys Ile Leu Ala Glu Phe Lys Ile Asp Glu Leu Ile
370 375 380
Lys Lys Leu Glu Lys Glu Leu Lys Lys Gly Asn Cys Asp Thr Glu Ile
385 390 395 400
Phe Gly Ile Phe Lys Lys His Tyr Lys Val Asn Phe Asp Ser Lys Lys
405 410 415
Phe Ser Lys Lys Ser Asp Glu Glu Lys Glu Leu Tyr Lys Ile Ile Tyr
420 425 430
Arg Tyr Leu Lys Gly Arg Ile Glu Lys Ile Leu Val Asn Glu Gln Lys
435 440 445
Val Arg Leu Lys Lys Met Glu Lys Ile Glu Ile Glu Lys Ile Leu Asn
450 455 460
Glu Ser Ile Leu Ser Glu Lys Ile Leu Lys Arg Val Lys Gln Tyr Thr
465 470 475 480
Leu Glu His Ile Met Tyr Leu Gly Lys Leu Arg His Asn Asp Ile Asp
485 490 495
Met Thr Thr Val Asn Thr Asp Asp Phe Ser Arg Leu His Ala Lys Glu
500 505 510
Glu Leu Asp Leu Glu Leu Ile Thr Phe Phe Ala Ser Thr Asn Met Glu
515 520 525
Leu Asn Lys Ile Phe Ser Arg Glu Asn Ile Asn Asn Asp Glu Asn Ile
530 535 540
Asp Phe Phe Gly Gly Asp Arg Glu Lys Asn Tyr Val Leu Asp Lys Lys
545 550 555 560
Ile Leu Asn Ser Lys Ile Lys Ile Ile Arg Asp Leu Asp Phe Ile Asp
565 570 575
Asn Lys Asn Asn Ile Thr Asn Asn Phe Ile Arg Lys Phe Thr Lys Ile
580 585 590
Gly Thr Asn Glu Arg Asn Arg Ile Leu His Ala Ile Ser Lys Glu Arg
595 600 605
Asp Leu Gln Gly Thr Gln Asp Asp Tyr Asn Lys Val Ile Asn Ile Ile
610 615 620
Gln Asn Leu Lys Ile Ser Asp Glu Glu Val Ser Lys Ala Leu Asn Leu
625 630 635 640
Asp Val Val Phe Lys Asp Lys Lys Asn Ile Ile Thr Lys Ile Asn Asp
645 650 655
Ile Lys Ile Ser Glu Glu Asn Asn Asn Asp Ile Lys Tyr Leu Pro Ser
660 665 670
Phe Ser Lys Val Leu Pro Glu Ile Leu Asn Leu Tyr Arg Asn Asn Pro
675 680 685
Lys Asn Glu Pro Phe Asp Thr Ile Glu Thr Glu Lys Ile Val Leu Asn
690 695 700
Ala Leu Ile Tyr Val Asn Lys Glu Leu Tyr Lys Lys Leu Ile Leu Glu
705 710 715 720
Asp Asp Leu Glu Glu Asn Glu Ser Lys Asn Ile Phe Leu Gln Glu Leu
725 730 735
Lys Lys Thr Leu Gly Asn Ile Asp Glu Ile Asp Glu Asn Ile Ile Glu
740 745 750
Asn Tyr Tyr Lys Asn Ala Gln Ile Ser Ala Ser Lys Gly Asn Asn Lys
755 760 765
Ala Ile Lys Lys Tyr Gln Lys Lys Val Ile Glu Cys Tyr Ile Gly Tyr
770 775 780
Leu Arg Lys Asn Tyr Glu Glu Leu Phe Asp Phe Ser Asp Phe Lys Met
785 790 795 800
Asn Ile Gln Glu Ile Lys Lys Gln Ile Lys Asp Ile Asn Asp Asn Lys
805 810 815
Thr Tyr Glu Arg Ile Thr Val Lys Thr Ser Asp Lys Thr Ile Val Ile
820 825 830
Asn Asp Asp Phe Glu Tyr Ile Ile Ser Ile Phe Ala Leu Leu Asn Ser
835 840 845
Asn Ala Val Ile Asn Lys Ile Arg Asn Arg Phe Phe Ala Thr Ser Val
850 855 860
Trp Leu Asn Thr Ser Glu Tyr Gln Asn Ile Ile Asp Ile Leu Asp Glu
865 870 875 880
Ile Met Gln Leu Asn Thr Leu Arg Asn Glu Cys Ile Thr Glu Asn Trp
885 890 895
Asn Leu Asn Leu Glu Glu Phe Ile Gln Lys Met Lys Glu Ile Glu Lys
900 905 910
Asp Phe Asp Asp Phe Lys Ile Gln Thr Lys Lys Glu Ile Phe Asn Asn
915 920 925
Tyr Tyr Glu Asp Ile Lys Asn Asn Ile Leu Thr Glu Phe Lys Asp Asp
930 935 940
Ile Asn Gly Cys Asp Val Leu Glu Lys Lys Leu Glu Lys Ile Val Ile
945 950 955 960
Phe Asp Asp Glu Thr Lys Phe Glu Ile Asp Lys Lys Ser Asn Ile Leu
965 970 975
Gln Asp Glu Gln Arg Lys Leu Ser Asn Ile Asn Lys Lys Asp Leu Lys
980 985 990
Lys Lys Val Asp Gln Tyr Ile Lys Asp Lys Asp Gln Glu Ile Lys Ser
995 1000 1005
Lys Ile Leu Cys Arg Ile Ile Phe Asn Ser Asp Phe Leu Lys Lys
1010 1015 1020
Tyr Lys Lys Glu Ile Asp Asn Leu Ile Glu Asp Met Glu Ser Glu
1025 1030 1035
Asn Glu Asn Lys Phe Gln Glu Ile Tyr Tyr Pro Lys Glu Arg Lys
1040 1045 1050
Asn Glu Leu Tyr Ile Tyr Lys Lys Asn Leu Phe Leu Asn Ile Gly
1055 1060 1065
Asn Pro Asn Phe Asp Lys Ile Tyr Gly Leu Ile Ser Asn Asp Ile
1070 1075 1080
Lys Met Ala Asp Ala Lys Phe Leu Phe Asn Ile Asp Gly Lys Asn
1085 1090 1095
Ile Arg Lys Asn Lys Ile Ser Glu Ile Asp Ala Ile Leu Lys Asn
1100 1105 1110
Leu Asn Asp Lys Leu Asn Gly Tyr Ser Lys Glu Tyr Lys Glu Lys
1115 1120 1125
Tyr Ile Lys Lys Leu Lys Glu Asn Asp Asp Phe Phe Ala Lys Asn
1130 1135 1140
Ile Gln Asn Lys Asn Tyr Lys Ser Phe Glu Lys Asp Tyr Asn Arg
1145 1150 1155
Val Ser Glu Tyr Lys Lys Ile Arg Asp Leu Val Glu Phe Asn Tyr
1160 1165 1170
Leu Asn Lys Ile Glu Ser Tyr Leu Ile Asp Ile Asn Trp Lys Leu
1175 1180 1185
Ala Ile Gln Met Ala Arg Phe Glu Arg Asp Met His Tyr Ile Val
1190 1195 1200
Asn Gly Leu Arg Glu Leu Gly Ile Ile Lys Leu Ser Gly Tyr Asn
1205 1210 1215
Thr Gly Ile Ser Arg Ala Tyr Pro Lys Arg Asn Gly Ser Asp Gly
1220 1225 1230
Phe Tyr Thr Thr Thr Ala Tyr Tyr Lys Phe Phe Asp Glu Glu Ser
1235 1240 1245
Tyr Lys Lys Phe Glu Lys Ile Cys Tyr Gly Phe Gly Ile Asp Leu
1250 1255 1260
Ser Glu Asn Ser Glu Ile Asn Lys Pro Glu Asn Glu Ser Ile Arg
1265 1270 1275
Asn Tyr Ile Ser His Phe Tyr Ile Val Arg Asn Pro Phe Ala Asp
1280 1285 1290
Tyr Ser Ile Ala Glu Gln Ile Asp Arg Val Ser Asn Leu Leu Ser
1295 1300 1305
Tyr Ser Thr Arg Tyr Asn Asn Ser Thr Tyr Ala Ser Val Phe Glu
1310 1315 1320
Val Phe Lys Lys Asp Val Asn Leu Asp Tyr Asp Glu Leu Lys Lys
1325 1330 1335
Lys Phe Lys Leu Ile Gly Asn Asn Asp Ile Leu Glu Arg Leu Met
1340 1345 1350
Lys Pro Lys Lys Val Ser Val Leu Glu Leu Glu Ser Tyr Asn Ser
1355 1360 1365
Asp Tyr Ile Lys Asn Leu Ile Ile Glu Leu Leu Thr Lys Ile Glu
1370 1375 1380
Asn Thr Asn Asp Thr Leu
1385
<210> 46
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 46
guuuuagucc ccuucgauau uggggugguc uauauc 36
<210> 47
<211> 95
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 47
auugaugugg uauacuaaaa auggaaaauu guauuuuuga uuagaaagau guaaaauuga 60
uuuaauuuaa aaauauuuua uuagauuaaa guaga 95
<210> 48
<211> 1300
<212> PRT
<213> Leptotrichia shahii
<400> 48
Met Ser Ile Tyr Gln Glu Phe Val Asn Lys Tyr Ser Leu Ser Lys Thr
1 5 10 15
Leu Arg Phe Glu Leu Ile Pro Gln Gly Lys Thr Leu Glu Asn Ile Lys
20 25 30
Ala Arg Gly Leu Ile Leu Asp Asp Glu Lys Arg Ala Lys Asp Tyr Lys
35 40 45
Lys Ala Lys Gln Ile Ile Asp Lys Tyr His Gln Phe Phe Ile Glu Glu
50 55 60
Ile Leu Ser Ser Val Cys Ile Ser Glu Asp Leu Leu Gln Asn Tyr Ser
65 70 75 80
Asp Val Tyr Phe Lys Leu Lys Lys Ser Asp Asp Asp Asn Leu Gln Lys
85 90 95
Asp Phe Lys Ser Ala Lys Asp Thr Ile Lys Lys Gln Ile Ser Glu Tyr
100 105 110
Ile Lys Asp Ser Glu Lys Phe Lys Asn Leu Phe Asn Gln Asn Leu Ile
115 120 125
Asp Ala Lys Lys Gly Gln Glu Ser Asp Leu Ile Leu Trp Leu Lys Gln
130 135 140
Ser Lys Asp Asn Gly Ile Glu Leu Phe Lys Ala Asn Ser Asp Ile Thr
145 150 155 160
Asp Ile Asp Glu Ala Leu Glu Ile Ile Lys Ser Phe Lys Gly Trp Thr
165 170 175
Thr Tyr Phe Lys Gly Phe His Glu Asn Arg Lys Asn Val Tyr Ser Ser
180 185 190
Asn Asp Ile Pro Thr Ser Ile Ile Tyr Arg Ile Val Asp Asp Asn Leu
195 200 205
Pro Lys Phe Leu Glu Asn Lys Ala Lys Tyr Glu Ser Leu Lys Asp Lys
210 215 220
Ala Pro Glu Ala Ile Asn Tyr Glu Gln Ile Lys Lys Asp Leu Ala Glu
225 230 235 240
Glu Leu Thr Phe Asp Ile Asp Tyr Lys Thr Ser Glu Val Asn Gln Arg
245 250 255
Val Phe Ser Leu Asp Glu Val Phe Glu Ile Ala Asn Phe Asn Asn Tyr
260 265 270
Leu Asn Gln Ser Gly Ile Thr Lys Phe Asn Thr Ile Ile Gly Gly Lys
275 280 285
Phe Val Asn Gly Glu Asn Thr Lys Arg Lys Gly Ile Asn Glu Tyr Ile
290 295 300
Asn Leu Tyr Ser Gln Gln Ile Asn Asp Lys Thr Leu Lys Lys Tyr Lys
305 310 315 320
Met Ser Val Leu Phe Lys Gln Ile Leu Ser Asp Thr Glu Ser Lys Ser
325 330 335
Phe Val Ile Asp Lys Leu Glu Asp Asp Ser Asp Val Val Thr Thr Met
340 345 350
Gln Ser Phe Tyr Glu Gln Ile Ala Ala Phe Lys Thr Val Glu Glu Lys
355 360 365
Ser Ile Lys Glu Thr Leu Ser Leu Leu Phe Asp Asp Leu Lys Ala Gln
370 375 380
Lys Leu Asp Leu Ser Lys Ile Tyr Phe Lys Asn Asp Lys Ser Leu Thr
385 390 395 400
Asp Leu Ser Gln Gln Val Phe Asp Asp Tyr Ser Val Ile Gly Thr Ala
405 410 415
Val Leu Glu Tyr Ile Thr Gln Gln Ile Ala Pro Lys Asn Leu Asp Asn
420 425 430
Pro Ser Lys Lys Glu Gln Glu Leu Ile Ala Lys Lys Thr Glu Lys Ala
435 440 445
Lys Tyr Leu Ser Leu Glu Thr Ile Lys Leu Ala Leu Glu Glu Phe Asn
450 455 460
Lys His Arg Asp Ile Asp Lys Gln Cys Arg Phe Glu Glu Ile Leu Ala
465 470 475 480
Asn Phe Ala Ala Ile Pro Met Ile Phe Asp Glu Ile Ala Gln Asn Lys
485 490 495
Asp Asn Leu Ala Gln Ile Ser Ile Lys Tyr Gln Asn Gln Gly Lys Lys
500 505 510
Asp Leu Leu Gln Ala Ser Ala Glu Asp Asp Val Lys Ala Ile Lys Asp
515 520 525
Leu Leu Asp Gln Thr Asn Asn Leu Leu His Lys Leu Lys Ile Phe His
530 535 540
Ile Ser Gln Ser Glu Asp Lys Ala Asn Ile Leu Asp Lys Asp Glu His
545 550 555 560
Phe Tyr Leu Val Phe Glu Glu Cys Tyr Phe Glu Leu Ala Asn Ile Val
565 570 575
Pro Leu Tyr Asn Lys Ile Arg Asn Tyr Ile Thr Gln Lys Pro Tyr Ser
580 585 590
Asp Glu Lys Phe Lys Leu Asn Phe Glu Asn Ser Thr Leu Ala Asn Gly
595 600 605
Trp Asp Lys Asn Lys Glu Pro Asp Asn Thr Ala Ile Leu Phe Ile Lys
610 615 620
Asp Asp Lys Tyr Tyr Leu Gly Val Met Asn Lys Lys Asn Asn Lys Ile
625 630 635 640
Phe Asp Asp Lys Ala Ile Lys Glu Asn Lys Gly Glu Gly Tyr Lys Lys
645 650 655
Ile Val Tyr Lys Leu Leu Pro Gly Ala Asn Lys Met Leu Pro Lys Val
660 665 670
Phe Phe Ser Ala Lys Ser Ile Lys Phe Tyr Asn Pro Ser Glu Asp Ile
675 680 685
Leu Arg Ile Arg Asn His Ser Thr His Thr Lys Asn Gly Ser Pro Gln
690 695 700
Lys Gly Tyr Glu Lys Phe Glu Phe Asn Ile Glu Asp Cys Arg Lys Phe
705 710 715 720
Ile Asp Phe Tyr Lys Gln Ser Ile Ser Lys His Pro Glu Trp Lys Asp
725 730 735
Phe Gly Phe Arg Phe Ser Asp Thr Gln Arg Tyr Asn Ser Ile Asp Glu
740 745 750
Phe Tyr Arg Glu Val Glu Asn Gln Gly Tyr Lys Leu Thr Phe Glu Asn
755 760 765
Ile Ser Glu Ser Tyr Ile Asp Ser Val Val Asn Gln Gly Lys Leu Tyr
770 775 780
Leu Phe Gln Ile Tyr Asn Lys Asp Phe Ser Ala Tyr Ser Lys Gly Arg
785 790 795 800
Pro Asn Leu His Thr Leu Tyr Trp Lys Ala Leu Phe Asp Glu Arg Asn
805 810 815
Leu Gln Asp Val Val Tyr Lys Leu Asn Gly Glu Ala Glu Leu Phe Tyr
820 825 830
Arg Lys Gln Ser Ile Pro Lys Lys Ile Thr His Pro Ala Lys Glu Ala
835 840 845
Ile Ala Asn Lys Asn Lys Asp Asn Pro Lys Lys Glu Ser Val Phe Glu
850 855 860
Tyr Asp Leu Ile Lys Asp Lys Arg Phe Thr Glu Asp Lys Phe Phe Phe
865 870 875 880
His Cys Pro Ile Thr Ile Asn Phe Lys Ser Ser Gly Ala Asn Lys Phe
885 890 895
Asn Asp Glu Ile Asn Leu Leu Leu Lys Glu Lys Ala Asn Asp Val His
900 905 910
Ile Leu Ser Ile Asp Arg Gly Glu Arg His Leu Ala Tyr Tyr Thr Leu
915 920 925
Val Asp Gly Lys Gly Asn Ile Ile Lys Gln Asp Thr Phe Asn Ile Ile
930 935 940
Gly Asn Asp Arg Met Lys Thr Asn Tyr His Asp Lys Leu Ala Ala Ile
945 950 955 960
Glu Lys Asp Arg Asp Ser Ala Arg Lys Asp Trp Lys Lys Ile Asn Asn
965 970 975
Ile Lys Glu Met Lys Glu Gly Tyr Leu Ser Gln Val Val His Glu Ile
980 985 990
Ala Lys Leu Val Ile Glu Tyr Asn Ala Ile Val Val Phe Glu Asp Leu
995 1000 1005
Asn Phe Gly Phe Lys Arg Gly Arg Phe Lys Val Glu Lys Gln Val
1010 1015 1020
Tyr Gln Lys Leu Glu Lys Met Leu Ile Glu Lys Leu Asn Tyr Leu
1025 1030 1035
Val Phe Lys Asp Asn Glu Phe Asp Lys Thr Gly Gly Val Leu Arg
1040 1045 1050
Ala Tyr Gln Leu Thr Ala Pro Phe Glu Thr Phe Lys Lys Met Gly
1055 1060 1065
Lys Gln Thr Gly Ile Ile Tyr Tyr Val Pro Ala Gly Phe Thr Ser
1070 1075 1080
Lys Ile Cys Pro Val Thr Gly Phe Val Asn Gln Leu Tyr Pro Lys
1085 1090 1095
Tyr Glu Ser Val Ser Lys Ser Gln Glu Phe Phe Ser Lys Phe Asp
1100 1105 1110
Lys Ile Cys Tyr Asn Leu Asp Lys Gly Tyr Phe Glu Phe Ser Phe
1115 1120 1125
Asp Tyr Lys Asn Phe Gly Asp Lys Ala Ala Lys Gly Lys Trp Thr
1130 1135 1140
Ile Ala Ser Phe Gly Ser Arg Leu Ile Asn Phe Arg Asn Ser Asp
1145 1150 1155
Lys Asn His Asn Trp Asp Thr Arg Glu Val Tyr Pro Thr Lys Glu
1160 1165 1170
Leu Glu Lys Leu Leu Lys Asp Tyr Ser Ile Glu Tyr Gly His Gly
1175 1180 1185
Glu Cys Ile Lys Ala Ala Ile Cys Gly Glu Ser Asp Lys Lys Phe
1190 1195 1200
Phe Ala Lys Leu Thr Ser Val Leu Asn Thr Ile Leu Gln Met Arg
1205 1210 1215
Asn Ser Lys Thr Gly Thr Glu Leu Asp Tyr Leu Ile Ser Pro Val
1220 1225 1230
Ala Asp Val Asn Gly Asn Phe Phe Asp Ser Arg Gln Ala Pro Lys
1235 1240 1245
Asn Met Pro Gln Asp Ala Asp Ala Asn Gly Ala Tyr His Ile Gly
1250 1255 1260
Leu Lys Gly Leu Met Leu Leu Gly Arg Ile Lys Asn Asn Gln Glu
1265 1270 1275
Gly Lys Lys Leu Asn Leu Val Ile Lys Asn Glu Glu Tyr Phe Glu
1280 1285 1290
Phe Val Gln Asn Arg Asn Asn
1295 1300
<210> 49
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 49
gucuaagaac uuuaaauaau uucuacuguu guagau 36
<210> 50
<211> 71
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 50
aucuacaaaa uuauaaacua aauaaagauu cuuauaauaa cuuuauauau aaucgaaaug 60
uagagaauuu u 71
<210> 51
<211> 1300
<212> PRT
<213> Francisella ularensis
<400> 51
Met Ser Ile Tyr Gln Glu Phe Val Asn Lys Tyr Ser Leu Ser Lys Thr
1 5 10 15
Leu Arg Phe Glu Leu Ile Pro Gln Gly Lys Thr Leu Glu Asn Ile Lys
20 25 30
Ala Arg Gly Leu Ile Leu Asp Asp Glu Lys Arg Ala Lys Asp Tyr Lys
35 40 45
Lys Ala Lys Gln Ile Ile Asp Lys Tyr His Gln Phe Phe Ile Glu Glu
50 55 60
Ile Leu Ser Ser Val Cys Ile Ser Glu Asp Leu Leu Gln Asn Tyr Ser
65 70 75 80
Asp Val Tyr Phe Lys Leu Lys Lys Ser Asp Asp Asp Asn Leu Gln Lys
85 90 95
Asp Phe Lys Ser Ala Lys Asp Thr Ile Lys Lys Gln Ile Ser Glu Tyr
100 105 110
Ile Lys Asp Ser Glu Lys Phe Lys Asn Leu Phe Asn Gln Asn Leu Ile
115 120 125
Asp Ala Lys Lys Gly Gln Glu Ser Asp Leu Ile Leu Trp Leu Lys Gln
130 135 140
Ser Lys Asp Asn Gly Ile Glu Leu Phe Lys Ala Asn Ser Asp Ile Thr
145 150 155 160
Asp Ile Asp Glu Ala Leu Glu Ile Ile Lys Ser Phe Lys Gly Trp Thr
165 170 175
Thr Tyr Phe Lys Gly Phe His Glu Asn Arg Lys Asn Val Tyr Ser Ser
180 185 190
Asn Asp Ile Pro Thr Ser Ile Ile Tyr Arg Ile Val Asp Asp Asn Leu
195 200 205
Pro Lys Phe Leu Glu Asn Lys Ala Lys Tyr Glu Ser Leu Lys Asp Lys
210 215 220
Ala Pro Glu Ala Ile Asn Tyr Glu Gln Ile Lys Lys Asp Leu Ala Glu
225 230 235 240
Glu Leu Thr Phe Asp Ile Asp Tyr Lys Thr Ser Glu Val Asn Gln Arg
245 250 255
Val Phe Ser Leu Asp Glu Val Phe Glu Ile Ala Asn Phe Asn Asn Tyr
260 265 270
Leu Asn Gln Ser Gly Ile Thr Lys Phe Asn Thr Ile Ile Gly Gly Lys
275 280 285
Phe Val Asn Gly Glu Asn Thr Lys Arg Lys Gly Ile Asn Glu Tyr Ile
290 295 300
Asn Leu Tyr Ser Gln Gln Ile Asn Asp Lys Thr Leu Lys Lys Tyr Lys
305 310 315 320
Met Ser Val Leu Phe Lys Gln Ile Leu Ser Asp Thr Glu Ser Lys Ser
325 330 335
Phe Val Ile Asp Lys Leu Glu Asp Asp Ser Asp Val Val Thr Thr Met
340 345 350
Gln Ser Phe Tyr Glu Gln Ile Ala Ala Phe Lys Thr Val Glu Glu Lys
355 360 365
Ser Ile Lys Glu Thr Leu Ser Leu Leu Phe Asp Asp Leu Lys Ala Gln
370 375 380
Lys Leu Asp Leu Ser Lys Ile Tyr Phe Lys Asn Asp Lys Ser Leu Thr
385 390 395 400
Asp Leu Ser Gln Gln Val Phe Asp Asp Tyr Ser Val Ile Gly Thr Ala
405 410 415
Val Leu Glu Tyr Ile Thr Gln Gln Ile Ala Pro Lys Asn Leu Asp Asn
420 425 430
Pro Ser Lys Lys Glu Gln Glu Leu Ile Ala Lys Lys Thr Glu Lys Ala
435 440 445
Lys Tyr Leu Ser Leu Glu Thr Ile Lys Leu Ala Leu Glu Glu Phe Asn
450 455 460
Lys His Arg Asp Ile Asp Lys Gln Cys Arg Phe Glu Glu Ile Leu Ala
465 470 475 480
Asn Phe Ala Ala Ile Pro Met Ile Phe Asp Glu Ile Ala Gln Asn Lys
485 490 495
Asp Asn Leu Ala Gln Ile Ser Ile Lys Tyr Gln Asn Gln Gly Lys Lys
500 505 510
Asp Leu Leu Gln Ala Ser Ala Glu Asp Asp Val Lys Ala Ile Lys Asp
515 520 525
Leu Leu Asp Gln Thr Asn Asn Leu Leu His Lys Leu Lys Ile Phe His
530 535 540
Ile Ser Gln Ser Glu Asp Lys Ala Asn Ile Leu Asp Lys Asp Glu His
545 550 555 560
Phe Tyr Leu Val Phe Glu Glu Cys Tyr Phe Glu Leu Ala Asn Ile Val
565 570 575
Pro Leu Tyr Asn Lys Ile Arg Asn Tyr Ile Thr Gln Lys Pro Tyr Ser
580 585 590
Asp Glu Lys Phe Lys Leu Asn Phe Glu Asn Ser Thr Leu Ala Asn Gly
595 600 605
Trp Asp Lys Asn Lys Glu Pro Asp Asn Thr Ala Ile Leu Phe Ile Lys
610 615 620
Asp Asp Lys Tyr Tyr Leu Gly Val Met Asn Lys Lys Asn Asn Lys Ile
625 630 635 640
Phe Asp Asp Lys Ala Ile Lys Glu Asn Lys Gly Glu Gly Tyr Lys Lys
645 650 655
Ile Val Tyr Lys Leu Leu Pro Gly Ala Asn Lys Met Leu Pro Lys Val
660 665 670
Phe Phe Ser Ala Lys Ser Ile Lys Phe Tyr Asn Pro Ser Glu Asp Ile
675 680 685
Leu Arg Ile Arg Asn His Ser Thr His Thr Lys Asn Gly Ser Pro Gln
690 695 700
Lys Gly Tyr Glu Lys Phe Glu Phe Asn Ile Glu Asp Cys Arg Lys Phe
705 710 715 720
Ile Asp Phe Tyr Lys Gln Ser Ile Ser Lys His Pro Glu Trp Lys Asp
725 730 735
Phe Gly Phe Arg Phe Ser Asp Thr Gln Arg Tyr Asn Ser Ile Asp Glu
740 745 750
Phe Tyr Arg Glu Val Glu Asn Gln Gly Tyr Lys Leu Thr Phe Glu Asn
755 760 765
Ile Ser Glu Ser Tyr Ile Asp Ser Val Val Asn Gln Gly Lys Leu Tyr
770 775 780
Leu Phe Gln Ile Tyr Asn Lys Asp Phe Ser Ala Tyr Ser Lys Gly Arg
785 790 795 800
Pro Asn Leu His Thr Leu Tyr Trp Lys Ala Leu Phe Asp Glu Arg Asn
805 810 815
Leu Gln Asp Val Val Tyr Lys Leu Asn Gly Glu Ala Glu Leu Phe Tyr
820 825 830
Arg Lys Gln Ser Ile Pro Lys Lys Ile Thr His Pro Ala Lys Glu Ala
835 840 845
Ile Ala Asn Lys Asn Lys Asp Asn Pro Lys Lys Glu Ser Val Phe Glu
850 855 860
Tyr Asp Leu Ile Lys Asp Lys Arg Phe Thr Glu Asp Lys Phe Phe Phe
865 870 875 880
His Cys Pro Ile Thr Ile Asn Phe Lys Ser Ser Gly Ala Asn Lys Phe
885 890 895
Asn Asp Glu Ile Asn Leu Leu Leu Lys Glu Lys Ala Asn Asp Val His
900 905 910
Ile Leu Ser Ile Asp Arg Gly Glu Arg His Leu Ala Tyr Tyr Thr Leu
915 920 925
Val Asp Gly Lys Gly Asn Ile Ile Lys Gln Asp Thr Phe Asn Ile Ile
930 935 940
Gly Asn Asp Arg Met Lys Thr Asn Tyr His Asp Lys Leu Ala Ala Ile
945 950 955 960
Glu Lys Asp Arg Asp Ser Ala Arg Lys Asp Trp Lys Lys Ile Asn Asn
965 970 975
Ile Lys Glu Met Lys Glu Gly Tyr Leu Ser Gln Val Val His Glu Ile
980 985 990
Ala Lys Leu Val Ile Glu Tyr Asn Ala Ile Val Val Phe Glu Asp Leu
995 1000 1005
Asn Phe Gly Phe Lys Arg Gly Arg Phe Lys Val Glu Lys Gln Val
1010 1015 1020
Tyr Gln Lys Leu Glu Lys Met Leu Ile Glu Lys Leu Asn Tyr Leu
1025 1030 1035
Val Phe Lys Asp Asn Glu Phe Asp Lys Thr Gly Gly Val Leu Arg
1040 1045 1050
Ala Tyr Gln Leu Thr Ala Pro Phe Glu Thr Phe Lys Lys Met Gly
1055 1060 1065
Lys Gln Thr Gly Ile Ile Tyr Tyr Val Pro Ala Gly Phe Thr Ser
1070 1075 1080
Lys Ile Cys Pro Val Thr Gly Phe Val Asn Gln Leu Tyr Pro Lys
1085 1090 1095
Tyr Glu Ser Val Ser Lys Ser Gln Glu Phe Phe Ser Lys Phe Asp
1100 1105 1110
Lys Ile Cys Tyr Asn Leu Asp Lys Gly Tyr Phe Glu Phe Ser Phe
1115 1120 1125
Asp Tyr Lys Asn Phe Gly Asp Lys Ala Ala Lys Gly Lys Trp Thr
1130 1135 1140
Ile Ala Ser Phe Gly Ser Arg Leu Ile Asn Phe Arg Asn Ser Asp
1145 1150 1155
Lys Asn His Asn Trp Asp Thr Arg Glu Val Tyr Pro Thr Lys Glu
1160 1165 1170
Leu Glu Lys Leu Leu Lys Asp Tyr Ser Ile Glu Tyr Gly His Gly
1175 1180 1185
Glu Cys Ile Lys Ala Ala Ile Cys Gly Glu Ser Asp Lys Lys Phe
1190 1195 1200
Phe Ala Lys Leu Thr Ser Val Leu Asn Thr Ile Leu Gln Met Arg
1205 1210 1215
Asn Ser Lys Thr Gly Thr Glu Leu Asp Tyr Leu Ile Ser Pro Val
1220 1225 1230
Ala Asp Val Asn Gly Asn Phe Phe Asp Ser Arg Gln Ala Pro Lys
1235 1240 1245
Asn Met Pro Gln Asp Ala Asp Ala Asn Gly Ala Tyr His Ile Gly
1250 1255 1260
Leu Lys Gly Leu Met Leu Leu Gly Arg Ile Lys Asn Asn Gln Glu
1265 1270 1275
Gly Lys Lys Leu Asn Leu Val Ile Lys Asn Glu Glu Tyr Phe Glu
1280 1285 1290
Phe Val Gln Asn Arg Asn Asn
1295 1300
<210> 52
<211> 138
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 52
aaaaggccgg cggccacgaa aaaggccggc caggcaaaaa agaaaaaggg atcctaccca 60
tacgatgttc cagattacgc ttatccctac gacgtgcctg attatgcata cccatatgat 120
gtccccgact atgcctaa 138
<210> 53
<211> 1388
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 53
Met Ser Leu Asn Arg Ile Tyr Gln Gly Arg Val Ala Ala Val Glu Thr
1 5 10 15
Gly Thr Ala Leu Ala Lys Gly Asn Val Glu Trp Met Pro Ala Ala Gly
20 25 30
Gly Asp Glu Val Leu Trp Gln His His Glu Leu Phe Gln Ala Ala Ile
35 40 45
Asn Tyr Tyr Leu Val Ala Leu Leu Ala Leu Ala Asp Lys Asn Asn Pro
50 55 60
Val Leu Gly Pro Leu Ile Ser Gln Met Asp Asn Pro Gln Ser Pro Tyr
65 70 75 80
His Val Trp Gly Ser Phe Arg Arg Gln Gly Arg Gln Arg Thr Gly Leu
85 90 95
Ser Gln Ala Val Ala Pro Tyr Ile Thr Pro Gly Asn Asn Ala Pro Thr
100 105 110
Leu Asp Glu Val Phe Arg Ser Ile Leu Ala Gly Asn Pro Thr Asp Arg
115 120 125
Ala Thr Leu Asp Ala Ala Leu Met Gln Leu Leu Lys Ala Cys Asp Gly
130 135 140
Ala Gly Ala Ile Gln Gln Glu Gly Arg Ser Tyr Trp Pro Lys Phe Cys
145 150 155 160
Asp Pro Asp Ser Thr Ala Asn Phe Ala Gly Asp Pro Ala Met Leu Arg
165 170 175
Arg Glu Gln His Arg Leu Leu Leu Pro Gln Val Leu His Asp Pro Ala
180 185 190
Ile Thr His Asp Ser Pro Ala Leu Gly Ser Phe Asp Thr Tyr Ser Ile
195 200 205
Ala Thr Pro Asp Thr Arg Thr Pro Gln Leu Thr Gly Pro Lys Ala Arg
210 215 220
Ala Arg Leu Glu Gln Ala Ile Thr Leu Trp Arg Val Arg Leu Pro Glu
225 230 235 240
Ser Ala Ala Asp Phe Asp Arg Leu Ala Ser Ser Leu Lys Lys Ile Pro
245 250 255
Asp Asp Asp Ser Arg Leu Asn Leu Gln Gly Tyr Val Gly Ser Ser Ala
260 265 270
Lys Gly Glu Val Gln Ala Arg Leu Phe Ala Leu Leu Leu Phe Arg His
275 280 285
Leu Glu Arg Ser Ser Phe Thr Leu Gly Leu Leu Arg Ser Ala Thr Pro
290 295 300
Pro Pro Lys Asn Ala Glu Thr Pro Pro Pro Ala Gly Val Pro Leu Pro
305 310 315 320
Ala Ala Ser Ala Ala Asp Pro Val Arg Ile Ala Arg Gly Lys Arg Ser
325 330 335
Phe Val Phe Arg Ala Phe Thr Ser Leu Pro Cys Trp His Gly Gly Asp
340 345 350
Asn Ile His Pro Thr Trp Lys Ser Phe Asp Ile Ala Ala Phe Lys Tyr
355 360 365
Ala Leu Thr Val Ile Asn Gln Ile Glu Glu Lys Thr Lys Glu Arg Gln
370 375 380
Lys Glu Cys Ala Glu Leu Glu Thr Asp Phe Asp Tyr Met His Gly Arg
385 390 395 400
Leu Ala Lys Ile Pro Val Lys Tyr Thr Thr Gly Glu Ala Glu Pro Pro
405 410 415
Pro Ile Leu Ala Asn Asp Leu Arg Ile Pro Leu Leu Arg Glu Leu Leu
420 425 430
Gln Asn Ile Lys Val Asp Thr Ala Leu Thr Asp Gly Glu Ala Val Ser
435 440 445
Tyr Gly Leu Gln Arg Arg Thr Ile Arg Gly Phe Arg Glu Leu Arg Arg
450 455 460
Ile Trp Arg Gly His Ala Pro Ala Gly Thr Val Phe Ser Ser Glu Leu
465 470 475 480
Lys Glu Lys Leu Ala Gly Glu Leu Arg Gln Phe Gln Thr Asp Asn Ser
485 490 495
Thr Thr Ile Gly Ser Val Gln Leu Phe Asn Glu Leu Ile Gln Asn Pro
500 505 510
Lys Tyr Trp Pro Ile Trp Gln Ala Pro Asp Val Glu Thr Ala Arg Gln
515 520 525
Trp Ala Asp Ala Gly Phe Ala Asp Asp Pro Leu Ala Ala Leu Val Gln
530 535 540
Glu Ala Glu Leu Gln Glu Asp Ile Asp Ala Leu Lys Ala Pro Val Lys
545 550 555 560
Leu Thr Pro Ala Asp Pro Glu Tyr Ser Arg Arg Gln Tyr Asp Phe Asn
565 570 575
Ala Val Ser Lys Phe Gly Ala Gly Ser Arg Ser Ala Asn Arg His Glu
580 585 590
Pro Gly Gln Thr Glu Arg Gly His Asn Thr Phe Thr Thr Glu Ile Ala
595 600 605
Ala Arg Asn Ala Ala Asp Gly Asn Arg Trp Arg Ala Thr His Val Arg
610 615 620
Ile His Tyr Ser Ala Pro Arg Leu Leu Arg Asp Gly Leu Arg Arg Pro
625 630 635 640
Asp Thr Asp Gly Asn Glu Ala Leu Glu Ala Val Pro Trp Leu Gln Pro
645 650 655
Met Met Glu Ala Leu Ala Pro Leu Pro Thr Leu Pro Gln Asp Leu Thr
660 665 670
Gly Met Pro Val Phe Leu Met Pro Asp Val Thr Leu Ser Gly Glu Arg
675 680 685
Arg Ile Leu Leu Asn Leu Pro Val Thr Leu Glu Pro Ala Ala Leu Val
690 695 700
Glu Gln Leu Gly Asn Ala Gly Arg Trp Gln Asn Gln Phe Phe Gly Ser
705 710 715 720
Arg Glu Asp Pro Phe Ala Leu Arg Trp Pro Ala Asp Gly Ala Val Lys
725 730 735
Thr Ala Lys Gly Lys Thr His Ile Pro Trp His Gln Asp Arg Asp His
740 745 750
Phe Thr Val Leu Gly Val Asp Leu Gly Thr Arg Asp Ala Gly Ala Leu
755 760 765
Ala Leu Leu Asn Val Thr Ala Gln Lys Pro Ala Lys Pro Val His Arg
770 775 780
Ile Ile Gly Glu Ala Asp Gly Arg Thr Trp Tyr Ala Ser Leu Ala Asp
785 790 795 800
Ala Arg Met Ile Arg Leu Pro Gly Glu Asp Ala Arg Leu Phe Val Arg
805 810 815
Gly Lys Leu Val Gln Glu Pro Tyr Gly Glu Arg Gly Arg Asn Ala Ser
820 825 830
Leu Leu Glu Trp Glu Asp Ala Arg Asn Ile Ile Leu Arg Leu Gly Gln
835 840 845
Asn Pro Asp Glu Leu Leu Gly Ala Asp Pro Arg Arg His Ser Tyr Pro
850 855 860
Glu Ile Asn Asp Lys Leu Leu Val Ala Leu Arg Arg Ala Gln Ala Arg
865 870 875 880
Leu Ala Arg Leu Gln Asn Arg Ser Trp Arg Leu Arg Asp Leu Ala Glu
885 890 895
Ser Asp Lys Ala Leu Asp Glu Ile His Ala Glu Arg Ala Gly Glu Lys
900 905 910
Pro Ser Pro Leu Pro Pro Leu Ala Arg Asp Asp Ala Ile Lys Ser Thr
915 920 925
Asp Glu Ala Leu Leu Ser Gln Arg Asp Ile Ile Arg Arg Ser Phe Val
930 935 940
Gln Ile Ala Asn Leu Ile Leu Pro Leu Arg Gly Arg Arg Trp Glu Trp
945 950 955 960
Arg Pro His Val Glu Val Pro Asp Cys His Ile Leu Ala Gln Ser Asp
965 970 975
Pro Gly Thr Asp Asp Thr Lys Arg Leu Val Ala Gly Gln Arg Gly Ile
980 985 990
Ser His Glu Arg Ile Glu Gln Ile Glu Glu Leu Arg Arg Arg Cys Gln
995 1000 1005
Ser Leu Asn Arg Ala Leu Arg His Lys Pro Gly Glu Arg Pro Val
1010 1015 1020
Leu Gly Arg Pro Ala Lys Gly Glu Glu Ile Ala Asp Pro Cys Pro
1025 1030 1035
Ala Leu Leu Glu Lys Ile Asn Arg Leu Arg Asp Gln Arg Val Asp
1040 1045 1050
Gln Thr Ala His Ala Ile Leu Ala Ala Ala Leu Gly Val Arg Leu
1055 1060 1065
Arg Ala Pro Ser Lys Asp Arg Ala Glu Arg Arg His Arg Asp Ile
1070 1075 1080
His Gly Glu Tyr Glu Arg Phe Arg Ala Pro Ala Asp Phe Val Val
1085 1090 1095
Ile Glu Asn Leu Ser Arg Tyr Leu Ser Ser Gln Asp Arg Ala Arg
1100 1105 1110
Ser Glu Asn Thr Arg Leu Met Gln Trp Cys His Arg Gln Ile Val
1115 1120 1125
Gln Lys Leu Arg Gln Leu Cys Glu Thr Tyr Gly Ile Pro Val Leu
1130 1135 1140
Ala Val Pro Ala Ala Tyr Ser Ser Arg Phe Ser Ser Arg Asp Gly
1145 1150 1155
Ser Ala Gly Phe Arg Ala Val His Leu Thr Pro Asp His Arg His
1160 1165 1170
Arg Met Pro Trp Ser Arg Ile Leu Ala Arg Leu Lys Ala His Glu
1175 1180 1185
Glu Asp Gly Lys Arg Leu Glu Lys Thr Val Leu Asp Glu Ala Arg
1190 1195 1200
Ala Val Arg Gly Leu Phe Asp Arg Leu Asp Arg Phe Asn Ala Gly
1205 1210 1215
His Val Pro Gly Lys Pro Trp Arg Thr Leu Leu Ala Pro Leu Pro
1220 1225 1230
Gly Gly Pro Val Phe Val Pro Leu Gly Asp Ala Thr Pro Met Gln
1235 1240 1245
Ala Asp Leu Asn Ala Ala Ile Asn Ile Ala Leu Arg Gly Ile Ala
1250 1255 1260
Ala Pro Asp Arg His Asp Ile His His Arg Leu Arg Ala Glu Asn
1265 1270 1275
Lys Lys Arg Ile Leu Ser Leu Arg Leu Gly Thr Gln Arg Glu Lys
1280 1285 1290
Ala Arg Trp Pro Gly Gly Ala Pro Ala Val Thr Leu Ser Thr Pro
1295 1300 1305
Asn Asn Gly Ala Ser Pro Glu Asp Ser Asp Ala Leu Pro Glu Arg
1310 1315 1320
Val Ser Asn Leu Phe Val Asp Ile Ala Gly Val Ala Asn Phe Glu
1325 1330 1335
Arg Val Thr Ile Glu Gly Val Ser Gln Lys Phe Ala Thr Gly Arg
1340 1345 1350
Gly Leu Trp Ala Ser Val Lys Gln Arg Ala Trp Asn Arg Val Ala
1355 1360 1365
Arg Leu Asn Glu Thr Val Thr Asp Asn Asn Arg Asn Glu Glu Glu
1370 1375 1380
Asp Asp Ile Pro Met
1385
<210> 54
<211> 1108
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 54
Met Ala Thr Arg Ser Phe Ile Leu Lys Ile Glu Pro Asn Glu Glu Val
1 5 10 15
Lys Lys Gly Leu Trp Lys Thr His Glu Val Leu Asn His Gly Ile Ala
20 25 30
Tyr Tyr Met Asn Ile Leu Lys Leu Ile Arg Gln Glu Ala Ile Tyr Glu
35 40 45
His His Glu Gln Asp Pro Lys Asn Pro Lys Lys Val Ser Lys Ala Glu
50 55 60
Ile Gln Ala Glu Leu Trp Asp Phe Val Leu Lys Met Gln Lys Cys Asn
65 70 75 80
Ser Phe Thr His Glu Val Asp Lys Asp Val Val Phe Asn Ile Leu Arg
85 90 95
Glu Leu Tyr Glu Glu Leu Val Pro Ser Ser Val Glu Lys Lys Gly Glu
100 105 110
Ala Asn Gln Leu Ser Asn Lys Phe Leu Tyr Pro Leu Val Asp Pro Asn
115 120 125
Ser Gln Ser Gly Lys Gly Thr Ala Ser Ser Gly Arg Lys Pro Arg Trp
130 135 140
Tyr Asn Leu Lys Ile Ala Gly Asp Pro Ser Trp Glu Glu Glu Lys Lys
145 150 155 160
Lys Trp Glu Glu Asp Lys Lys Lys Asp Pro Leu Ala Lys Ile Leu Gly
165 170 175
Lys Leu Ala Glu Tyr Gly Leu Ile Pro Leu Phe Ile Pro Phe Thr Asp
180 185 190
Ser Asn Glu Pro Ile Val Lys Glu Ile Lys Trp Met Glu Lys Ser Arg
195 200 205
Asn Gln Ser Val Arg Arg Leu Asp Lys Asp Met Phe Ile Gln Ala Leu
210 215 220
Glu Arg Phe Leu Ser Trp Glu Ser Trp Asn Leu Lys Val Lys Glu Glu
225 230 235 240
Tyr Glu Lys Val Glu Lys Glu His Lys Thr Leu Glu Glu Arg Ile Lys
245 250 255
Glu Asp Ile Gln Ala Phe Lys Ser Leu Glu Gln Tyr Glu Lys Glu Arg
260 265 270
Gln Glu Gln Leu Leu Arg Asp Thr Leu Asn Thr Asn Glu Tyr Arg Leu
275 280 285
Ser Lys Arg Gly Leu Arg Gly Trp Arg Glu Ile Ile Gln Lys Trp Leu
290 295 300
Lys Met Asp Glu Asn Glu Pro Ser Glu Lys Tyr Leu Glu Val Phe Lys
305 310 315 320
Asp Tyr Gln Arg Lys His Pro Arg Glu Ala Gly Asp Tyr Ser Val Tyr
325 330 335
Glu Phe Leu Ser Lys Lys Glu Asn His Phe Ile Trp Arg Asn His Pro
340 345 350
Glu Tyr Pro Tyr Leu Tyr Ala Thr Phe Cys Glu Ile Asp Lys Lys Lys
355 360 365
Lys Asp Ala Lys Gln Gln Ala Thr Phe Thr Leu Ala Asp Pro Ile Asn
370 375 380
His Pro Leu Trp Val Arg Phe Glu Glu Arg Ser Gly Ser Asn Leu Asn
385 390 395 400
Lys Tyr Arg Ile Leu Thr Glu Gln Leu His Thr Glu Lys Leu Lys Lys
405 410 415
Lys Leu Thr Val Gln Leu Asp Arg Leu Ile Tyr Pro Thr Glu Ser Gly
420 425 430
Gly Trp Glu Glu Lys Gly Lys Val Asp Ile Val Leu Leu Pro Ser Arg
435 440 445
Gln Phe Tyr Asn Gln Ile Phe Leu Asp Ile Glu Glu Lys Gly Lys His
450 455 460
Ala Phe Thr Tyr Lys Asp Glu Ser Ile Lys Phe Pro Leu Lys Gly Thr
465 470 475 480
Leu Gly Gly Ala Arg Val Gln Phe Asp Arg Asp His Leu Arg Arg Tyr
485 490 495
Pro His Lys Val Glu Ser Gly Asn Val Gly Arg Ile Tyr Phe Asn Met
500 505 510
Thr Val Asn Ile Glu Pro Thr Glu Ser Pro Val Ser Lys Ser Leu Lys
515 520 525
Ile His Arg Asp Asp Phe Pro Lys Phe Val Asn Phe Lys Pro Lys Glu
530 535 540
Leu Thr Glu Trp Ile Lys Asp Ser Lys Gly Lys Lys Leu Lys Ser Gly
545 550 555 560
Ile Glu Ser Leu Glu Ile Gly Leu Arg Val Met Ser Ile Asp Leu Gly
565 570 575
Gln Arg Gln Ala Ala Ala Ala Ser Ile Phe Glu Val Val Asp Gln Lys
580 585 590
Pro Asp Ile Glu Gly Lys Leu Phe Phe Pro Ile Lys Gly Thr Glu Leu
595 600 605
Tyr Ala Val His Arg Ala Ser Phe Asn Ile Lys Leu Pro Gly Glu Thr
610 615 620
Leu Val Lys Ser Arg Glu Val Leu Arg Lys Ala Arg Glu Asp Asn Leu
625 630 635 640
Lys Leu Met Asn Gln Lys Leu Asn Phe Leu Arg Asn Val Leu His Phe
645 650 655
Gln Gln Phe Glu Asp Ile Thr Glu Arg Glu Lys Arg Val Thr Lys Trp
660 665 670
Ile Ser Arg Gln Glu Asn Ser Asp Val Pro Leu Val Tyr Gln Asp Glu
675 680 685
Leu Ile Gln Ile Arg Glu Leu Met Tyr Lys Pro Tyr Lys Asp Trp Val
690 695 700
Ala Phe Leu Lys Gln Leu His Lys Arg Leu Glu Val Glu Ile Gly Lys
705 710 715 720
Glu Val Lys His Trp Arg Lys Ser Leu Ser Asp Gly Arg Lys Gly Leu
725 730 735
Tyr Gly Ile Ser Leu Lys Asn Ile Asp Glu Ile Asp Arg Thr Arg Lys
740 745 750
Phe Leu Leu Arg Trp Ser Leu Arg Pro Thr Glu Pro Gly Glu Val Arg
755 760 765
Arg Leu Glu Pro Gly Gln Arg Phe Ala Ile Asp Gln Leu Asn His Leu
770 775 780
Asn Ala Leu Lys Glu Asp Arg Leu Lys Lys Met Ala Asn Thr Ile Ile
785 790 795 800
Met His Ala Leu Gly Tyr Cys Tyr Asp Val Arg Lys Lys Lys Trp Gln
805 810 815
Ala Lys Asn Pro Ala Cys Gln Ile Ile Leu Phe Glu Asp Leu Ser Asn
820 825 830
Tyr Asn Pro Tyr Glu Glu Arg Ser Arg Phe Glu Asn Ser Lys Leu Met
835 840 845
Lys Trp Ser Arg Arg Glu Ile Pro Arg Gln Val Ala Leu Gln Gly Glu
850 855 860
Ile Tyr Gly Leu Gln Val Gly Glu Val Gly Ala Gln Phe Ser Ser Arg
865 870 875 880
Phe His Ala Lys Thr Gly Ser Pro Gly Ile Arg Cys Ser Val Val Thr
885 890 895
Lys Glu Lys Leu Gln Asp Asn Arg Phe Phe Lys Asn Leu Gln Arg Glu
900 905 910
Gly Arg Leu Thr Leu Asp Lys Ile Ala Val Leu Lys Glu Gly Asp Leu
915 920 925
Tyr Pro Asp Lys Gly Gly Glu Lys Phe Ile Ser Leu Ser Lys Asp Arg
930 935 940
Lys Leu Val Thr Thr His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln
945 950 955 960
Lys Arg Phe Trp Thr Arg Thr His Gly Phe Tyr Lys Val Tyr Cys Lys
965 970 975
Ala Tyr Gln Val Asp Gly Gln Thr Val Tyr Ile Pro Glu Ser Lys Asp
980 985 990
Gln Lys Gln Lys Ile Ile Glu Glu Phe Gly Glu Gly Tyr Phe Ile Leu
995 1000 1005
Lys Asp Gly Val Tyr Glu Trp Gly Asn Ala Gly Lys Leu Lys Ile
1010 1015 1020
Lys Lys Gly Ser Ser Lys Gln Ser Ser Ser Glu Leu Val Asp Ser
1025 1030 1035
Asp Ile Leu Lys Asp Ser Phe Asp Leu Ala Ser Glu Leu Lys Gly
1040 1045 1050
Glu Lys Leu Met Leu Tyr Arg Asp Pro Ser Gly Asn Val Phe Pro
1055 1060 1065
Ser Asp Lys Trp Met Ala Ala Gly Val Phe Phe Gly Lys Leu Glu
1070 1075 1080
Arg Ile Leu Ile Ser Lys Leu Thr Asn Gln Tyr Ser Ile Ser Thr
1085 1090 1095
Ile Glu Asp Asp Ser Ser Lys Gln Ser Met
1100 1105
<210> 55
<211> 1108
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 55
Met Ala Ile Arg Ser Ile Lys Leu Lys Leu Lys Thr His Thr Gly Pro
1 5 10 15
Glu Ala Gln Asn Leu Arg Lys Gly Ile Trp Arg Thr His Arg Leu Leu
20 25 30
Asn Glu Gly Val Ala Tyr Tyr Met Lys Met Leu Leu Leu Phe Arg Gln
35 40 45
Glu Ser Thr Gly Glu Arg Pro Lys Glu Glu Leu Gln Glu Glu Leu Ile
50 55 60
Cys His Ile Arg Glu Gln Gln Gln Arg Asn Gln Ala Asp Lys Asn Thr
65 70 75 80
Gln Ala Leu Pro Leu Asp Lys Ala Leu Glu Ala Leu Arg Gln Leu Tyr
85 90 95
Glu Leu Leu Val Pro Ser Ser Val Gly Gln Ser Gly Asp Ala Gln Ile
100 105 110
Ile Ser Arg Lys Phe Leu Ser Pro Leu Val Asp Pro Asn Ser Glu Gly
115 120 125
Gly Lys Gly Thr Ser Lys Ala Gly Ala Lys Pro Thr Trp Gln Lys Lys
130 135 140
Lys Glu Ala Asn Asp Pro Thr Trp Glu Gln Asp Tyr Glu Lys Trp Lys
145 150 155 160
Lys Arg Arg Glu Glu Asp Pro Thr Ala Ser Val Ile Thr Thr Leu Glu
165 170 175
Glu Tyr Gly Ile Arg Pro Ile Phe Pro Leu Tyr Thr Asn Thr Val Thr
180 185 190
Asp Ile Ala Trp Leu Pro Leu Gln Ser Asn Gln Phe Val Arg Thr Trp
195 200 205
Asp Arg Asp Met Leu Gln Gln Ala Ile Glu Arg Leu Leu Ser Trp Glu
210 215 220
Ser Trp Asn Lys Arg Val Gln Glu Glu Tyr Ala Lys Leu Lys Glu Lys
225 230 235 240
Met Ala Gln Leu Asn Glu Gln Leu Glu Gly Gly Gln Glu Trp Ile Ser
245 250 255
Leu Leu Glu Gln Tyr Glu Glu Asn Arg Glu Arg Glu Leu Arg Glu Asn
260 265 270
Met Thr Ala Ala Asn Asp Lys Tyr Arg Ile Thr Lys Arg Gln Met Lys
275 280 285
Gly Trp Asn Glu Leu Tyr Glu Leu Trp Ser Thr Phe Pro Ala Ser Ala
290 295 300
Ser His Glu Gln Tyr Lys Glu Ala Leu Lys Arg Val Gln Gln Arg Leu
305 310 315 320
Arg Gly Arg Phe Gly Asp Ala His Phe Phe Gln Tyr Leu Met Glu Glu
325 330 335
Lys Asn Arg Leu Ile Trp Lys Gly Asn Pro Gln Arg Ile His Tyr Phe
340 345 350
Val Ala Arg Asn Glu Leu Thr Lys Arg Leu Glu Glu Ala Lys Gln Ser
355 360 365
Ala Thr Met Thr Leu Pro Asn Ala Arg Lys His Pro Leu Trp Val Arg
370 375 380
Phe Asp Ala Arg Gly Gly Asn Leu Gln Asp Tyr Tyr Leu Thr Ala Glu
385 390 395 400
Ala Asp Lys Pro Arg Ser Arg Arg Phe Val Thr Phe Ser Gln Leu Ile
405 410 415
Trp Pro Ser Glu Ser Gly Trp Met Glu Lys Lys Asp Val Glu Val Glu
420 425 430
Leu Ala Leu Ser Arg Gln Phe Tyr Gln Gln Val Lys Leu Leu Lys Asn
435 440 445
Asp Lys Gly Lys Gln Lys Ile Glu Phe Lys Asp Lys Gly Ser Gly Ser
450 455 460
Thr Phe Asn Gly His Leu Gly Gly Ala Lys Leu Gln Leu Glu Arg Gly
465 470 475 480
Asp Leu Glu Lys Glu Glu Lys Asn Phe Glu Asp Gly Glu Ile Gly Ser
485 490 495
Val Tyr Leu Asn Val Val Ile Asp Phe Glu Pro Leu Gln Glu Val Lys
500 505 510
Asn Gly Arg Val Gln Ala Pro Tyr Gly Gln Val Leu Gln Leu Ile Arg
515 520 525
Arg Pro Asn Glu Phe Pro Lys Val Thr Thr Tyr Lys Ser Glu Gln Leu
530 535 540
Val Glu Trp Ile Lys Ala Ser Pro Gln His Ser Ala Gly Val Glu Ser
545 550 555 560
Leu Ala Ser Gly Phe Arg Val Met Ser Ile Asp Leu Gly Leu Arg Ala
565 570 575
Ala Ala Ala Thr Ser Ile Phe Ser Val Glu Glu Ser Ser Asp Lys Asn
580 585 590
Ala Ala Asp Phe Ser Tyr Trp Ile Glu Gly Thr Pro Leu Val Ala Val
595 600 605
His Gln Arg Ser Tyr Met Leu Arg Leu Pro Gly Glu Gln Val Glu Lys
610 615 620
Gln Val Met Glu Lys Arg Asp Glu Arg Phe Gln Leu His Gln Arg Val
625 630 635 640
Lys Phe Gln Ile Arg Val Leu Ala Gln Ile Met Arg Met Ala Asn Lys
645 650 655
Gln Tyr Gly Asp Arg Trp Asp Glu Leu Asp Ser Leu Lys Gln Ala Val
660 665 670
Glu Gln Lys Lys Ser Pro Leu Asp Gln Thr Asp Arg Thr Phe Trp Glu
675 680 685
Gly Ile Val Cys Asp Leu Thr Lys Val Leu Pro Arg Asn Glu Ala Asp
690 695 700
Trp Glu Gln Ala Val Val Gln Ile His Arg Lys Ala Glu Glu Tyr Val
705 710 715 720
Gly Lys Ala Val Gln Ala Trp Arg Lys Arg Phe Ala Ala Asp Glu Arg
725 730 735
Lys Gly Ile Ala Gly Leu Ser Met Trp Asn Ile Glu Glu Leu Glu Gly
740 745 750
Leu Arg Lys Leu Leu Ile Ser Trp Ser Arg Arg Thr Arg Asn Pro Gln
755 760 765
Glu Val Asn Arg Phe Glu Arg Gly His Thr Ser His Gln Arg Leu Leu
770 775 780
Thr His Ile Gln Asn Val Lys Glu Asp Arg Leu Lys Gln Leu Ser His
785 790 795 800
Ala Ile Val Met Thr Ala Leu Gly Tyr Val Tyr Asp Glu Arg Lys Gln
805 810 815
Glu Trp Cys Ala Glu Tyr Pro Ala Cys Gln Val Ile Leu Phe Glu Asn
820 825 830
Leu Ser Gln Tyr Arg Ser Asn Leu Asp Arg Ser Thr Lys Glu Asn Ser
835 840 845
Thr Leu Met Lys Trp Ala His Arg Ser Ile Pro Lys Tyr Val His Met
850 855 860
Gln Ala Glu Pro Tyr Gly Ile Gln Ile Gly Asp Val Arg Ala Glu Tyr
865 870 875 880
Ser Ser Arg Phe Tyr Ala Lys Thr Gly Thr Pro Gly Ile Arg Cys Lys
885 890 895
Lys Val Arg Gly Gln Asp Leu Gln Gly Arg Arg Phe Glu Asn Leu Gln
900 905 910
Lys Arg Leu Val Asn Glu Gln Phe Leu Thr Glu Glu Gln Val Lys Gln
915 920 925
Leu Arg Pro Gly Asp Ile Val Pro Asp Asp Ser Gly Glu Leu Phe Met
930 935 940
Thr Leu Thr Asp Gly Ser Gly Ser Lys Glu Val Val Phe Leu Gln Ala
945 950 955 960
Asp Ile Asn Ala Ala His Asn Leu Gln Lys Arg Phe Trp Gln Arg Tyr
965 970 975
Asn Glu Leu Phe Lys Val Ser Cys Arg Val Ile Val Arg Asp Glu Glu
980 985 990
Glu Tyr Leu Val Pro Lys Thr Lys Ser Val Gln Ala Lys Leu Gly Lys
995 1000 1005
Gly Leu Phe Val Lys Lys Ser Asp Thr Ala Trp Lys Asp Val Tyr
1010 1015 1020
Val Trp Asp Ser Gln Ala Lys Leu Lys Gly Lys Thr Thr Phe Thr
1025 1030 1035
Glu Glu Ser Glu Ser Pro Glu Gln Leu Glu Asp Phe Gln Glu Ile
1040 1045 1050
Ile Glu Glu Ala Glu Glu Ala Lys Gly Thr Tyr Arg Thr Leu Phe
1055 1060 1065
Arg Asp Pro Ser Gly Val Phe Phe Pro Glu Ser Val Trp Tyr Pro
1070 1075 1080
Gln Lys Asp Phe Trp Gly Glu Val Lys Arg Lys Leu Tyr Gly Lys
1085 1090 1095
Leu Arg Glu Arg Phe Leu Thr Lys Ala Arg
1100 1105
<210> 56
<211> 1334
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 56
Met Lys Ile Ser Lys Val Asp His Thr Arg Met Ala Val Ala Lys Gly
1 5 10 15
Asn Gln His Arg Arg Asp Glu Ile Ser Gly Ile Leu Tyr Lys Asp Pro
20 25 30
Thr Lys Thr Gly Ser Ile Asp Phe Asp Glu Arg Phe Lys Lys Leu Asn
35 40 45
Cys Ser Ala Lys Ile Leu Tyr His Val Phe Asn Gly Ile Ala Glu Gly
50 55 60
Ser Asn Lys Tyr Lys Asn Ile Val Asp Lys Val Asn Asn Asn Leu Asp
65 70 75 80
Arg Val Leu Phe Thr Gly Lys Ser Tyr Asp Arg Lys Ser Ile Ile Asp
85 90 95
Ile Asp Thr Val Leu Arg Asn Val Glu Lys Ile Asn Ala Phe Asp Arg
100 105 110
Ile Ser Thr Glu Glu Arg Glu Gln Ile Ile Asp Asp Leu Leu Glu Ile
115 120 125
Gln Leu Arg Lys Gly Leu Arg Lys Gly Lys Ala Gly Leu Arg Glu Val
130 135 140
Leu Leu Ile Gly Ala Gly Val Ile Val Arg Thr Asp Lys Lys Gln Glu
145 150 155 160
Ile Ala Asp Phe Leu Glu Ile Leu Asp Glu Asp Phe Asn Lys Thr Asn
165 170 175
Gln Ala Lys Asn Ile Lys Leu Ser Ile Glu Asn Gln Gly Leu Val Val
180 185 190
Ser Pro Val Ser Arg Gly Glu Glu Arg Ile Phe Asp Val Ser Gly Ala
195 200 205
Gln Lys Gly Lys Ser Ser Lys Lys Ala Gln Glu Lys Glu Ala Leu Ser
210 215 220
Ala Phe Leu Leu Asp Tyr Ala Asp Leu Asp Lys Asn Val Arg Phe Glu
225 230 235 240
Tyr Leu Arg Lys Ile Arg Arg Leu Ile Asn Leu Tyr Phe Tyr Val Lys
245 250 255
Asn Asp Asp Val Met Ser Leu Thr Glu Ile Pro Ala Glu Val Asn Leu
260 265 270
Glu Lys Asp Phe Asp Ile Trp Arg Asp His Glu Gln Arg Lys Glu Glu
275 280 285
Asn Gly Asp Phe Val Gly Cys Pro Asp Ile Leu Leu Ala Asp Arg Asp
290 295 300
Val Lys Lys Ser Asn Ser Lys Gln Val Lys Ile Ala Glu Arg Gln Leu
305 310 315 320
Arg Glu Ser Ile Arg Glu Lys Asn Ile Lys Arg Tyr Arg Phe Ser Ile
325 330 335
Lys Thr Ile Glu Lys Asp Asp Gly Thr Tyr Phe Phe Ala Asn Lys Gln
340 345 350
Ile Ser Val Phe Trp Ile His Arg Ile Glu Asn Ala Val Glu Arg Ile
355 360 365
Leu Gly Ser Ile Asn Asp Lys Lys Leu Tyr Arg Leu Arg Leu Gly Tyr
370 375 380
Leu Gly Glu Lys Val Trp Lys Asp Ile Leu Asn Phe Leu Ser Ile Lys
385 390 395 400
Tyr Ile Ala Val Gly Lys Ala Val Phe Asn Phe Ala Met Asp Asp Leu
405 410 415
Gln Glu Lys Asp Arg Asp Ile Glu Pro Gly Lys Ile Ser Glu Asn Ala
420 425 430
Val Asn Gly Leu Thr Ser Phe Asp Tyr Glu Gln Ile Lys Ala Asp Glu
435 440 445
Met Leu Gln Arg Glu Val Ala Val Asn Val Ala Phe Ala Ala Asn Asn
450 455 460
Leu Ala Arg Val Thr Val Asp Ile Pro Gln Asn Gly Glu Lys Glu Asp
465 470 475 480
Ile Leu Leu Trp Asn Lys Ser Asp Ile Lys Lys Tyr Lys Lys Asn Ser
485 490 495
Lys Lys Gly Ile Leu Lys Ser Ile Leu Gln Phe Phe Gly Gly Ala Ser
500 505 510
Thr Trp Asn Met Lys Met Phe Glu Ile Ala Tyr His Asp Gln Pro Gly
515 520 525
Asp Tyr Glu Glu Asn Tyr Leu Tyr Asp Ile Ile Gln Ile Ile Tyr Ser
530 535 540
Leu Arg Asn Lys Ser Phe His Phe Lys Thr Tyr Asp His Gly Asp Lys
545 550 555 560
Asn Trp Asn Arg Glu Leu Ile Gly Lys Met Ile Glu His Asp Ala Glu
565 570 575
Arg Val Ile Ser Val Glu Arg Glu Lys Phe His Ser Asn Asn Leu Pro
580 585 590
Met Phe Tyr Lys Asp Ala Asp Leu Lys Lys Ile Leu Asp Leu Leu Tyr
595 600 605
Ser Asp Tyr Ala Gly Arg Ala Ser Gln Val Pro Ala Phe Asn Thr Val
610 615 620
Leu Val Arg Lys Asn Phe Pro Glu Phe Leu Arg Lys Asp Met Gly Tyr
625 630 635 640
Lys Val His Phe Asn Asn Pro Glu Val Glu Asn Gln Trp His Ser Ala
645 650 655
Val Tyr Tyr Leu Tyr Lys Glu Ile Tyr Tyr Asn Leu Phe Leu Arg Asp
660 665 670
Lys Glu Val Lys Asn Leu Phe Tyr Thr Ser Leu Lys Asn Ile Arg Ser
675 680 685
Glu Val Ser Asp Lys Lys Gln Lys Leu Ala Ser Asp Asp Phe Ala Ser
690 695 700
Arg Cys Glu Glu Ile Glu Asp Arg Ser Leu Pro Glu Ile Cys Gln Ile
705 710 715 720
Ile Met Thr Glu Tyr Asn Ala Gln Asn Phe Gly Asn Arg Lys Val Lys
725 730 735
Ser Gln Arg Val Ile Glu Lys Asn Lys Asp Ile Phe Arg His Tyr Lys
740 745 750
Met Leu Leu Ile Lys Thr Leu Ala Gly Ala Phe Ser Leu Tyr Leu Lys
755 760 765
Gln Glu Arg Phe Ala Phe Ile Gly Lys Ala Thr Pro Ile Pro Tyr Glu
770 775 780
Thr Thr Asp Val Lys Asn Phe Leu Pro Glu Trp Lys Ser Gly Met Tyr
785 790 795 800
Ala Ser Phe Val Glu Glu Ile Lys Asn Asn Leu Asp Leu Gln Glu Trp
805 810 815
Tyr Ile Val Gly Arg Phe Leu Asn Gly Arg Met Leu Asn Gln Leu Ala
820 825 830
Gly Ser Leu Arg Ser Tyr Ile Gln Tyr Ala Glu Asp Ile Glu Arg Arg
835 840 845
Ala Ala Glu Asn Arg Asn Lys Leu Phe Ser Lys Pro Asp Glu Lys Ile
850 855 860
Glu Ala Cys Lys Lys Ala Val Arg Val Leu Asp Leu Cys Ile Lys Ile
865 870 875 880
Ser Thr Arg Ile Ser Ala Glu Phe Thr Asp Tyr Phe Asp Ser Glu Asp
885 890 895
Asp Tyr Ala Asp Tyr Leu Glu Lys Tyr Leu Lys Tyr Gln Asp Asp Ala
900 905 910
Ile Lys Glu Leu Ser Gly Ser Ser Tyr Ala Ala Leu Asp His Phe Cys
915 920 925
Asn Lys Asp Asp Leu Lys Phe Asp Ile Tyr Val Asn Ala Gly Gln Lys
930 935 940
Pro Ile Leu Gln Arg Asn Ile Val Met Ala Lys Leu Phe Gly Pro Asp
945 950 955 960
Asn Ile Leu Ser Glu Val Met Glu Lys Val Thr Glu Ser Ala Ile Arg
965 970 975
Glu Tyr Tyr Asp Tyr Leu Lys Lys Val Ser Gly Tyr Arg Val Arg Gly
980 985 990
Lys Cys Ser Thr Glu Lys Glu Gln Glu Asp Leu Leu Lys Phe Gln Arg
995 1000 1005
Leu Lys Asn Ala Val Glu Phe Arg Asp Val Thr Glu Tyr Ala Glu
1010 1015 1020
Val Ile Asn Glu Leu Leu Gly Gln Leu Ile Ser Trp Ser Tyr Leu
1025 1030 1035
Arg Glu Arg Asp Leu Leu Tyr Phe Gln Leu Gly Phe His Tyr Met
1040 1045 1050
Cys Leu Lys Asn Lys Ser Phe Lys Pro Ala Glu Tyr Val Asp Ile
1055 1060 1065
Arg Arg Asn Asn Gly Thr Ile Ile His Asn Ala Ile Leu Tyr Gln
1070 1075 1080
Ile Val Ser Met Tyr Ile Asn Gly Leu Asp Phe Tyr Ser Cys Asp
1085 1090 1095
Lys Glu Gly Lys Thr Leu Lys Pro Ile Glu Thr Gly Lys Gly Val
1100 1105 1110
Gly Ser Lys Ile Gly Gln Phe Ile Lys Tyr Ser Gln Tyr Leu Tyr
1115 1120 1125
Asn Asp Pro Ser Tyr Lys Leu Glu Ile Tyr Asn Ala Gly Leu Glu
1130 1135 1140
Val Phe Glu Asn Ile Asp Glu His Asp Asn Ile Thr Asp Leu Arg
1145 1150 1155
Lys Tyr Val Asp His Phe Lys Tyr Tyr Ala Tyr Gly Asn Lys Met
1160 1165 1170
Ser Leu Leu Asp Leu Tyr Ser Glu Phe Phe Asp Arg Phe Phe Thr
1175 1180 1185
Tyr Asp Met Lys Tyr Gln Lys Asn Val Val Asn Val Leu Glu Asn
1190 1195 1200
Ile Leu Leu Arg His Phe Val Ile Phe Tyr Pro Lys Phe Gly Ser
1205 1210 1215
Gly Lys Lys Asp Val Gly Ile Arg Asp Cys Lys Lys Glu Arg Ala
1220 1225 1230
Gln Ile Glu Ile Ser Glu Gln Ser Leu Thr Ser Glu Asp Phe Met
1235 1240 1245
Phe Lys Leu Asp Asp Lys Ala Gly Glu Glu Ala Lys Lys Phe Pro
1250 1255 1260
Ala Arg Asp Glu Arg Tyr Leu Gln Thr Ile Ala Lys Leu Leu Tyr
1265 1270 1275
Tyr Pro Asn Glu Ile Glu Asp Met Asn Arg Phe Met Lys Lys Gly
1280 1285 1290
Glu Thr Ile Asn Lys Lys Val Gln Phe Asn Arg Lys Lys Lys Ile
1295 1300 1305
Thr Arg Lys Gln Lys Asn Asn Ser Ser Asn Glu Val Leu Ser Ser
1310 1315 1320
Thr Met Gly Tyr Leu Phe Lys Asn Ile Lys Leu
1325 1330
<210> 57
<211> 1120
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 57
Met Trp Ile Ser Ile Lys Thr Leu Ile His His Leu Gly Val Leu Phe
1 5 10 15
Phe Cys Asp Tyr Met Tyr Asn Arg Arg Glu Lys Lys Ile Ile Glu Val
20 25 30
Lys Thr Met Arg Ile Thr Lys Val Glu Val Asp Arg Lys Lys Val Leu
35 40 45
Ile Ser Arg Asp Lys Asn Gly Gly Lys Leu Val Tyr Glu Asn Glu Met
50 55 60
Gln Asp Asn Thr Glu Gln Ile Met His His Lys Lys Ser Ser Phe Tyr
65 70 75 80
Lys Ser Val Val Asn Lys Thr Ile Cys Arg Pro Glu Gln Lys Gln Met
85 90 95
Lys Lys Leu Val His Gly Leu Leu Gln Glu Asn Ser Gln Glu Lys Ile
100 105 110
Lys Val Ser Asp Val Thr Lys Leu Asn Ile Ser Asn Phe Leu Asn His
115 120 125
Arg Phe Lys Lys Ser Leu Tyr Tyr Phe Pro Glu Asn Ser Pro Asp Lys
130 135 140
Ser Glu Glu Tyr Arg Ile Glu Ile Asn Leu Ser Gln Leu Leu Glu Asp
145 150 155 160
Ser Leu Lys Lys Gln Gln Gly Thr Phe Ile Cys Trp Glu Ser Phe Ser
165 170 175
Lys Asp Met Glu Leu Tyr Ile Asn Trp Ala Glu Asn Tyr Ile Ser Ser
180 185 190
Lys Thr Lys Leu Ile Lys Lys Ser Ile Arg Asn Asn Arg Ile Gln Ser
195 200 205
Thr Glu Ser Arg Ser Gly Gln Leu Met Asp Arg Tyr Met Lys Asp Ile
210 215 220
Leu Asn Lys Asn Lys Pro Phe Asp Ile Gln Ser Val Ser Glu Lys Tyr
225 230 235 240
Gln Leu Glu Lys Leu Thr Ser Ala Leu Lys Ala Thr Phe Lys Glu Ala
245 250 255
Lys Lys Asn Asp Lys Glu Ile Asn Tyr Lys Leu Lys Ser Thr Leu Gln
260 265 270
Asn His Glu Arg Gln Ile Ile Glu Glu Leu Lys Glu Asn Ser Glu Leu
275 280 285
Asn Gln Phe Asn Ile Glu Ile Arg Lys His Leu Glu Thr Tyr Phe Pro
290 295 300
Ile Lys Lys Thr Asn Arg Lys Val Gly Asp Ile Arg Asn Leu Glu Ile
305 310 315 320
Gly Glu Ile Gln Lys Ile Val Asn His Arg Leu Lys Asn Lys Ile Val
325 330 335
Gln Arg Ile Leu Gln Glu Gly Lys Leu Ala Ser Tyr Glu Ile Glu Ser
340 345 350
Thr Val Asn Ser Asn Ser Leu Gln Lys Ile Lys Ile Glu Glu Ala Phe
355 360 365
Ala Leu Lys Phe Ile Asn Ala Cys Leu Phe Ala Ser Asn Asn Leu Arg
370 375 380
Asn Met Val Tyr Pro Val Cys Lys Lys Asp Ile Leu Met Ile Gly Glu
385 390 395 400
Phe Lys Asn Ser Phe Lys Glu Ile Lys His Lys Lys Phe Ile Arg Gln
405 410 415
Trp Ser Gln Phe Phe Ser Gln Glu Ile Thr Val Asp Asp Ile Glu Leu
420 425 430
Ala Ser Trp Gly Leu Arg Gly Ala Ile Ala Pro Ile Arg Asn Glu Ile
435 440 445
Ile His Leu Lys Lys His Ser Trp Lys Lys Phe Phe Asn Asn Pro Thr
450 455 460
Phe Lys Val Lys Lys Ser Lys Ile Ile Asn Gly Lys Thr Lys Asp Val
465 470 475 480
Thr Ser Glu Phe Leu Tyr Lys Glu Thr Leu Phe Lys Asp Tyr Phe Tyr
485 490 495
Ser Glu Leu Asp Ser Val Pro Glu Leu Ile Ile Asn Lys Met Glu Ser
500 505 510
Ser Lys Ile Leu Asp Tyr Tyr Ser Ser Asp Gln Leu Asn Gln Val Phe
515 520 525
Thr Ile Pro Asn Phe Glu Leu Ser Leu Leu Thr Ser Ala Val Pro Phe
530 535 540
Ala Pro Ser Phe Lys Arg Val Tyr Leu Lys Gly Phe Asp Tyr Gln Asn
545 550 555 560
Gln Asp Glu Ala Gln Pro Asp Tyr Asn Leu Lys Leu Asn Ile Tyr Asn
565 570 575
Glu Lys Ala Phe Asn Ser Glu Ala Phe Gln Ala Gln Tyr Ser Leu Phe
580 585 590
Lys Met Val Tyr Tyr Gln Val Phe Leu Pro Gln Phe Thr Thr Asn Asn
595 600 605
Asp Leu Phe Lys Ser Ser Val Asp Phe Ile Leu Thr Leu Asn Lys Glu
610 615 620
Arg Lys Gly Tyr Ala Lys Ala Phe Gln Asp Ile Arg Lys Met Asn Lys
625 630 635 640
Asp Glu Lys Pro Ser Glu Tyr Met Ser Tyr Ile Gln Ser Gln Leu Met
645 650 655
Leu Tyr Gln Lys Lys Gln Glu Glu Lys Glu Lys Ile Asn His Phe Glu
660 665 670
Lys Phe Ile Asn Gln Val Phe Ile Lys Gly Phe Asn Ser Phe Ile Glu
675 680 685
Lys Asn Arg Leu Thr Tyr Ile Cys His Pro Thr Lys Asn Thr Val Pro
690 695 700
Glu Asn Asp Asn Ile Glu Ile Pro Phe His Thr Asp Met Asp Asp Ser
705 710 715 720
Asn Ile Ala Phe Trp Leu Met Cys Lys Leu Leu Asp Ala Lys Gln Leu
725 730 735
Ser Glu Leu Arg Asn Glu Met Ile Lys Phe Ser Cys Ser Leu Gln Ser
740 745 750
Thr Glu Glu Ile Ser Thr Phe Thr Lys Ala Arg Glu Val Ile Gly Leu
755 760 765
Ala Leu Leu Asn Gly Glu Lys Gly Cys Asn Asp Trp Lys Glu Leu Phe
770 775 780
Asp Asp Lys Glu Ala Trp Lys Lys Asn Met Ser Leu Tyr Val Ser Glu
785 790 795 800
Glu Leu Leu Gln Ser Leu Pro Tyr Thr Gln Glu Asp Gly Gln Thr Pro
805 810 815
Val Ile Asn Arg Ser Ile Asp Leu Val Lys Lys Tyr Gly Thr Glu Thr
820 825 830
Ile Leu Glu Lys Leu Phe Ser Ser Ser Asp Asp Tyr Lys Val Ser Ala
835 840 845
Lys Asp Ile Ala Lys Leu His Glu Tyr Asp Val Thr Glu Lys Ile Ala
850 855 860
Gln Gln Glu Ser Leu His Lys Gln Trp Ile Glu Lys Pro Gly Leu Ala
865 870 875 880
Arg Asp Ser Ala Trp Thr Lys Lys Tyr Gln Asn Val Ile Asn Asp Ile
885 890 895
Ser Asn Tyr Gln Trp Ala Lys Thr Lys Val Glu Leu Thr Gln Val Arg
900 905 910
His Leu His Gln Leu Thr Ile Asp Leu Leu Ser Arg Leu Ala Gly Tyr
915 920 925
Met Ser Ile Ala Asp Arg Asp Phe Gln Phe Ser Ser Asn Tyr Ile Leu
930 935 940
Glu Arg Glu Asn Ser Glu Tyr Arg Val Thr Ser Trp Ile Leu Leu Ser
945 950 955 960
Glu Asn Lys Asn Lys Asn Lys Tyr Asn Asp Tyr Glu Leu Tyr Asn Leu
965 970 975
Lys Asn Ala Ser Ile Lys Val Ser Ser Lys Asn Asp Pro Gln Leu Lys
980 985 990
Val Asp Leu Lys Gln Leu Arg Leu Thr Leu Glu Tyr Leu Glu Leu Phe
995 1000 1005
Asp Asn Arg Leu Lys Glu Lys Arg Asn Asn Ile Ser His Phe Asn
1010 1015 1020
Tyr Leu Asn Gly Gln Leu Gly Asn Ser Ile Leu Glu Leu Phe Asp
1025 1030 1035
Asp Ala Arg Asp Val Leu Ser Tyr Asp Arg Lys Leu Lys Asn Ala
1040 1045 1050
Val Ser Lys Ser Leu Lys Glu Ile Leu Ser Ser His Gly Met Glu
1055 1060 1065
Val Thr Phe Lys Pro Leu Tyr Gln Thr Asn His His Leu Lys Ile
1070 1075 1080
Asp Lys Leu Gln Pro Lys Lys Ile His His Leu Gly Glu Lys Ser
1085 1090 1095
Thr Val Ser Ser Asn Gln Val Ser Asn Glu Tyr Cys Gln Leu Val
1100 1105 1110
Arg Thr Leu Leu Thr Met Lys
1115 1120
<210> 58
<211> 1152
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 58
Met Lys Val Thr Lys Val Asp Gly Ile Ser His Lys Lys Tyr Ile Glu
1 5 10 15
Glu Gly Lys Leu Val Lys Ser Thr Ser Glu Glu Asn Arg Thr Ser Glu
20 25 30
Arg Leu Ser Glu Leu Leu Ser Ile Arg Leu Asp Ile Tyr Ile Lys Asn
35 40 45
Pro Asp Asn Ala Ser Glu Glu Glu Asn Arg Ile Arg Arg Glu Asn Leu
50 55 60
Lys Lys Phe Phe Ser Asn Lys Val Leu His Leu Lys Asp Ser Val Leu
65 70 75 80
Tyr Leu Lys Asn Arg Lys Glu Lys Asn Ala Val Gln Asp Lys Asn Tyr
85 90 95
Ser Glu Glu Asp Ile Ser Glu Tyr Asp Leu Lys Asn Lys Asn Ser Phe
100 105 110
Ser Val Leu Lys Lys Ile Leu Leu Asn Glu Asp Val Asn Ser Glu Glu
115 120 125
Leu Glu Ile Phe Arg Lys Asp Val Glu Ala Lys Leu Asn Lys Ile Asn
130 135 140
Ser Leu Lys Tyr Ser Phe Glu Glu Asn Lys Ala Asn Tyr Gln Lys Ile
145 150 155 160
Asn Glu Asn Asn Val Glu Lys Val Gly Gly Lys Ser Lys Arg Asn Ile
165 170 175
Ile Tyr Asp Tyr Tyr Arg Glu Ser Ala Lys Arg Asn Asp Tyr Ile Asn
180 185 190
Asn Val Gln Glu Ala Phe Asp Lys Leu Tyr Lys Lys Glu Asp Ile Glu
195 200 205
Lys Leu Phe Phe Leu Ile Glu Asn Ser Lys Lys His Glu Lys Tyr Lys
210 215 220
Ile Arg Glu Tyr Tyr His Lys Ile Ile Gly Arg Lys Asn Asp Lys Glu
225 230 235 240
Asn Phe Ala Lys Ile Ile Tyr Glu Glu Ile Gln Asn Val Asn Asn Ile
245 250 255
Lys Glu Leu Ile Glu Lys Ile Pro Asp Met Ser Glu Leu Lys Lys Ser
260 265 270
Gln Val Phe Tyr Lys Tyr Tyr Leu Asp Lys Glu Glu Leu Asn Asp Lys
275 280 285
Asn Ile Lys Tyr Ala Phe Cys His Phe Val Glu Ile Glu Met Ser Gln
290 295 300
Leu Leu Lys Asn Tyr Val Tyr Lys Arg Leu Ser Asn Ile Ser Asn Asp
305 310 315 320
Lys Ile Lys Arg Ile Phe Glu Tyr Gln Asn Leu Lys Lys Leu Ile Glu
325 330 335
Asn Lys Leu Leu Asn Lys Leu Asp Thr Tyr Val Arg Asn Cys Gly Lys
340 345 350
Tyr Asn Tyr Tyr Leu Gln Val Gly Glu Ile Ala Thr Ser Asp Phe Ile
355 360 365
Ala Arg Asn Arg Gln Asn Glu Ala Phe Leu Arg Asn Ile Ile Gly Val
370 375 380
Ser Ser Val Ala Tyr Phe Ser Leu Arg Asn Ile Leu Glu Thr Glu Asn
385 390 395 400
Glu Asn Asp Ile Thr Gly Arg Met Arg Gly Lys Thr Val Lys Asn Asn
405 410 415
Lys Gly Glu Glu Lys Tyr Val Ser Gly Glu Val Asp Lys Ile Tyr Asn
420 425 430
Glu Asn Lys Gln Asn Glu Val Lys Glu Asn Leu Lys Met Phe Tyr Ser
435 440 445
Tyr Asp Phe Asn Met Asp Asn Lys Asn Glu Ile Glu Asp Phe Phe Ala
450 455 460
Asn Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe
465 470 475 480
Asn Leu Glu Leu Glu Gly Lys Asp Ile Phe Ala Phe Lys Asn Ile Ala
485 490 495
Pro Ser Glu Ile Ser Lys Lys Met Phe Gln Asn Glu Ile Asn Glu Lys
500 505 510
Lys Leu Lys Leu Lys Ile Phe Lys Gln Leu Asn Ser Ala Asn Val Phe
515 520 525
Asn Tyr Tyr Glu Lys Asp Val Ile Ile Lys Tyr Leu Lys Asn Thr Lys
530 535 540
Phe Asn Phe Val Asn Lys Asn Ile Pro Phe Val Pro Ser Phe Thr Lys
545 550 555 560
Leu Tyr Asn Lys Ile Glu Asp Leu Arg Asn Thr Leu Lys Phe Phe Trp
565 570 575
Ser Val Pro Lys Asp Lys Glu Glu Lys Asp Ala Gln Ile Tyr Leu Leu
580 585 590
Lys Asn Ile Tyr Tyr Gly Glu Phe Leu Asn Lys Phe Val Lys Asn Ser
595 600 605
Lys Val Phe Phe Lys Ile Thr Asn Glu Val Ile Lys Ile Asn Lys Gln
610 615 620
Arg Asn Gln Lys Thr Gly His Tyr Lys Tyr Gln Lys Phe Glu Asn Ile
625 630 635 640
Glu Lys Thr Val Pro Val Glu Tyr Leu Ala Ile Ile Gln Ser Arg Glu
645 650 655
Met Ile Asn Asn Gln Asp Lys Glu Glu Lys Asn Thr Tyr Ile Asp Phe
660 665 670
Ile Gln Gln Ile Phe Leu Lys Gly Phe Ile Asp Tyr Leu Asn Lys Asn
675 680 685
Asn Leu Lys Tyr Ile Glu Ser Asn Asn Asn Asn Asp Asn Asn Asp Ile
690 695 700
Phe Ser Lys Ile Lys Ile Lys Lys Asp Asn Lys Glu Lys Tyr Asp Lys
705 710 715 720
Ile Leu Lys Asn Tyr Glu Lys His Asn Arg Asn Lys Glu Ile Pro His
725 730 735
Glu Ile Asn Glu Phe Val Arg Glu Ile Lys Leu Gly Lys Ile Leu Lys
740 745 750
Tyr Thr Glu Asn Leu Asn Met Phe Tyr Leu Ile Leu Lys Leu Leu Asn
755 760 765
His Lys Glu Leu Thr Asn Leu Lys Gly Ser Leu Glu Lys Tyr Gln Ser
770 775 780
Ala Asn Lys Glu Glu Thr Phe Ser Asp Glu Leu Glu Leu Ile Asn Leu
785 790 795 800
Leu Asn Leu Asp Asn Asn Arg Val Thr Glu Asp Phe Glu Leu Glu Ala
805 810 815
Asn Glu Ile Gly Lys Phe Leu Asp Phe Asn Glu Asn Lys Ile Lys Asp
820 825 830
Arg Lys Glu Leu Lys Lys Phe Asp Thr Asn Lys Ile Tyr Phe Asp Gly
835 840 845
Glu Asn Ile Ile Lys His Arg Ala Phe Tyr Asn Ile Lys Lys Tyr Gly
850 855 860
Met Leu Asn Leu Leu Glu Lys Ile Ala Asp Lys Ala Lys Tyr Lys Ile
865 870 875 880
Ser Leu Lys Glu Leu Lys Glu Tyr Ser Asn Lys Lys Asn Glu Ile Glu
885 890 895
Lys Asn Tyr Thr Met Gln Gln Asn Leu His Arg Lys Tyr Ala Arg Pro
900 905 910
Lys Lys Asp Glu Lys Phe Asn Asp Glu Asp Tyr Lys Glu Tyr Glu Lys
915 920 925
Ala Ile Gly Asn Ile Gln Lys Tyr Thr His Leu Lys Asn Lys Val Glu
930 935 940
Phe Asn Glu Leu Asn Leu Leu Gln Gly Leu Leu Leu Lys Ile Leu His
945 950 955 960
Arg Leu Val Gly Tyr Thr Ser Ile Trp Glu Arg Asp Leu Arg Phe Arg
965 970 975
Leu Lys Gly Glu Phe Pro Glu Asn His Tyr Ile Glu Glu Ile Phe Asn
980 985 990
Phe Asp Asn Ser Lys Asn Val Lys Tyr Lys Ser Gly Gln Ile Val Glu
995 1000 1005
Lys Tyr Ile Asn Phe Tyr Lys Glu Leu Tyr Lys Asp Asn Val Glu
1010 1015 1020
Lys Arg Ser Ile Tyr Ser Asp Lys Lys Val Lys Lys Leu Lys Gln
1025 1030 1035
Glu Lys Lys Asp Leu Tyr Ile Arg Asn Tyr Ile Ala His Phe Asn
1040 1045 1050
Tyr Ile Pro His Ala Glu Ile Ser Leu Leu Glu Val Leu Glu Asn
1055 1060 1065
Leu Arg Lys Leu Leu Ser Tyr Asp Arg Lys Leu Lys Asn Ala Ile
1070 1075 1080
Met Lys Ser Ile Val Asp Ile Leu Lys Glu Tyr Gly Phe Val Ala
1085 1090 1095
Thr Phe Lys Ile Gly Ala Asp Lys Lys Ile Glu Ile Gln Thr Leu
1100 1105 1110
Glu Ser Glu Lys Ile Val His Leu Lys Asn Leu Lys Lys Lys Lys
1115 1120 1125
Leu Met Thr Asp Arg Asn Ser Glu Glu Leu Cys Glu Leu Val Lys
1130 1135 1140
Val Met Phe Glu Tyr Lys Ala Leu Glu
1145 1150
<210> 59
<211> 1389
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 59
Met Gly Asn Leu Phe Gly His Lys Arg Trp Tyr Glu Val Arg Asp Lys
1 5 10 15
Lys Asp Phe Lys Ile Lys Arg Lys Val Lys Val Lys Arg Asn Tyr Asp
20 25 30
Gly Asn Lys Tyr Ile Leu Asn Ile Asn Glu Asn Asn Asn Lys Glu Lys
35 40 45
Ile Asp Asn Asn Lys Phe Ile Arg Lys Tyr Ile Asn Tyr Lys Lys Asn
50 55 60
Asp Asn Ile Leu Lys Glu Phe Thr Arg Lys Phe His Ala Gly Asn Ile
65 70 75 80
Leu Phe Lys Leu Lys Gly Lys Glu Gly Ile Ile Arg Ile Glu Asn Asn
85 90 95
Asp Asp Phe Leu Glu Thr Glu Glu Val Val Leu Tyr Ile Glu Ala Tyr
100 105 110
Gly Lys Ser Glu Lys Leu Lys Ala Leu Gly Ile Thr Lys Lys Lys Ile
115 120 125
Ile Asp Glu Ala Ile Arg Gln Gly Ile Thr Lys Asp Asp Lys Lys Ile
130 135 140
Glu Ile Lys Arg Gln Glu Asn Glu Glu Glu Ile Glu Ile Asp Ile Arg
145 150 155 160
Asp Glu Tyr Thr Asn Lys Thr Leu Asn Asp Cys Ser Ile Ile Leu Arg
165 170 175
Ile Ile Glu Asn Asp Glu Leu Glu Thr Lys Lys Ser Ile Tyr Glu Ile
180 185 190
Phe Lys Asn Ile Asn Met Ser Leu Tyr Lys Ile Ile Glu Lys Ile Ile
195 200 205
Glu Asn Glu Thr Glu Lys Val Phe Glu Asn Arg Tyr Tyr Glu Glu His
210 215 220
Leu Arg Glu Lys Leu Leu Lys Asp Asp Lys Ile Asp Val Ile Leu Thr
225 230 235 240
Asn Phe Met Glu Ile Arg Glu Lys Ile Lys Ser Asn Leu Glu Ile Leu
245 250 255
Gly Phe Val Lys Phe Tyr Leu Asn Val Gly Gly Asp Lys Lys Lys Ser
260 265 270
Lys Asn Lys Lys Met Leu Val Glu Lys Ile Leu Asn Ile Asn Val Asp
275 280 285
Leu Thr Val Glu Asp Ile Ala Asp Phe Val Ile Lys Glu Leu Glu Phe
290 295 300
Trp Asn Ile Thr Lys Arg Ile Glu Lys Val Lys Lys Val Asn Asn Glu
305 310 315 320
Phe Leu Glu Lys Arg Arg Asn Arg Thr Tyr Ile Lys Ser Tyr Val Leu
325 330 335
Leu Asp Lys His Glu Lys Phe Lys Ile Glu Arg Glu Asn Lys Lys Asp
340 345 350
Lys Ile Val Lys Phe Phe Val Glu Asn Ile Lys Asn Asn Ser Ile Lys
355 360 365
Glu Lys Ile Glu Lys Ile Leu Ala Glu Phe Lys Ile Asp Glu Leu Ile
370 375 380
Lys Lys Leu Glu Lys Glu Leu Lys Lys Gly Asn Cys Asp Thr Glu Ile
385 390 395 400
Phe Gly Ile Phe Lys Lys His Tyr Lys Val Asn Phe Asp Ser Lys Lys
405 410 415
Phe Ser Lys Lys Ser Asp Glu Glu Lys Glu Leu Tyr Lys Ile Ile Tyr
420 425 430
Arg Tyr Leu Lys Gly Arg Ile Glu Lys Ile Leu Val Asn Glu Gln Lys
435 440 445
Val Arg Leu Lys Lys Met Glu Lys Ile Glu Ile Glu Lys Ile Leu Asn
450 455 460
Glu Ser Ile Leu Ser Glu Lys Ile Leu Lys Arg Val Lys Gln Tyr Thr
465 470 475 480
Leu Glu His Ile Met Tyr Leu Gly Lys Leu Arg His Asn Asp Ile Asp
485 490 495
Met Thr Thr Val Asn Thr Asp Asp Phe Ser Arg Leu His Ala Lys Glu
500 505 510
Glu Leu Asp Leu Glu Leu Ile Thr Phe Phe Ala Ser Thr Asn Met Glu
515 520 525
Leu Asn Lys Ile Phe Ser Arg Glu Asn Ile Asn Asn Asp Glu Asn Ile
530 535 540
Asp Phe Phe Gly Gly Asp Arg Glu Lys Asn Tyr Val Leu Asp Lys Lys
545 550 555 560
Ile Leu Asn Ser Lys Ile Lys Ile Ile Arg Asp Leu Asp Phe Ile Asp
565 570 575
Asn Lys Asn Asn Ile Thr Asn Asn Phe Ile Arg Lys Phe Thr Lys Ile
580 585 590
Gly Thr Asn Glu Arg Asn Arg Ile Leu His Ala Ile Ser Lys Glu Arg
595 600 605
Asp Leu Gln Gly Thr Gln Asp Asp Tyr Asn Lys Val Ile Asn Ile Ile
610 615 620
Gln Asn Leu Lys Ile Ser Asp Glu Glu Val Ser Lys Ala Leu Asn Leu
625 630 635 640
Asp Val Val Phe Lys Asp Lys Lys Asn Ile Ile Thr Lys Ile Asn Asp
645 650 655
Ile Lys Ile Ser Glu Glu Asn Asn Asn Asp Ile Lys Tyr Leu Pro Ser
660 665 670
Phe Ser Lys Val Leu Pro Glu Ile Leu Asn Leu Tyr Arg Asn Asn Pro
675 680 685
Lys Asn Glu Pro Phe Asp Thr Ile Glu Thr Glu Lys Ile Val Leu Asn
690 695 700
Ala Leu Ile Tyr Val Asn Lys Glu Leu Tyr Lys Lys Leu Ile Leu Glu
705 710 715 720
Asp Asp Leu Glu Glu Asn Glu Ser Lys Asn Ile Phe Leu Gln Glu Leu
725 730 735
Lys Lys Thr Leu Gly Asn Ile Asp Glu Ile Asp Glu Asn Ile Ile Glu
740 745 750
Asn Tyr Tyr Lys Asn Ala Gln Ile Ser Ala Ser Lys Gly Asn Asn Lys
755 760 765
Ala Ile Lys Lys Tyr Gln Lys Lys Val Ile Glu Cys Tyr Ile Gly Tyr
770 775 780
Leu Arg Lys Asn Tyr Glu Glu Leu Phe Asp Phe Ser Asp Phe Lys Met
785 790 795 800
Asn Ile Gln Glu Ile Lys Lys Gln Ile Lys Asp Ile Asn Asp Asn Lys
805 810 815
Thr Tyr Glu Arg Ile Thr Val Lys Thr Ser Asp Lys Thr Ile Val Ile
820 825 830
Asn Asp Asp Phe Glu Tyr Ile Ile Ser Ile Phe Ala Leu Leu Asn Ser
835 840 845
Asn Ala Val Ile Asn Lys Ile Arg Asn Arg Phe Phe Ala Thr Ser Val
850 855 860
Trp Leu Asn Thr Ser Glu Tyr Gln Asn Ile Ile Asp Ile Leu Asp Glu
865 870 875 880
Ile Met Gln Leu Asn Thr Leu Arg Asn Glu Cys Ile Thr Glu Asn Trp
885 890 895
Asn Leu Asn Leu Glu Glu Phe Ile Gln Lys Met Lys Glu Ile Glu Lys
900 905 910
Asp Phe Asp Asp Phe Lys Ile Gln Thr Lys Lys Glu Ile Phe Asn Asn
915 920 925
Tyr Tyr Glu Asp Ile Lys Asn Asn Ile Leu Thr Glu Phe Lys Asp Asp
930 935 940
Ile Asn Gly Cys Asp Val Leu Glu Lys Lys Leu Glu Lys Ile Val Ile
945 950 955 960
Phe Asp Asp Glu Thr Lys Phe Glu Ile Asp Lys Lys Ser Asn Ile Leu
965 970 975
Gln Asp Glu Gln Arg Lys Leu Ser Asn Ile Asn Lys Lys Asp Leu Lys
980 985 990
Lys Lys Val Asp Gln Tyr Ile Lys Asp Lys Asp Gln Glu Ile Lys Ser
995 1000 1005
Lys Ile Leu Cys Arg Ile Ile Phe Asn Ser Asp Phe Leu Lys Lys
1010 1015 1020
Tyr Lys Lys Glu Ile Asp Asn Leu Ile Glu Asp Met Glu Ser Glu
1025 1030 1035
Asn Glu Asn Lys Phe Gln Glu Ile Tyr Tyr Pro Lys Glu Arg Lys
1040 1045 1050
Asn Glu Leu Tyr Ile Tyr Lys Lys Asn Leu Phe Leu Asn Ile Gly
1055 1060 1065
Asn Pro Asn Phe Asp Lys Ile Tyr Gly Leu Ile Ser Asn Asp Ile
1070 1075 1080
Lys Met Ala Asp Ala Lys Phe Leu Phe Asn Ile Asp Gly Lys Asn
1085 1090 1095
Ile Arg Lys Asn Lys Ile Ser Glu Ile Asp Ala Ile Leu Lys Asn
1100 1105 1110
Leu Asn Asp Lys Leu Asn Gly Tyr Ser Lys Glu Tyr Lys Glu Lys
1115 1120 1125
Tyr Ile Lys Lys Leu Lys Glu Asn Asp Asp Phe Phe Ala Lys Asn
1130 1135 1140
Ile Gln Asn Lys Asn Tyr Lys Ser Phe Glu Lys Asp Tyr Asn Arg
1145 1150 1155
Val Ser Glu Tyr Lys Lys Ile Arg Asp Leu Val Glu Phe Asn Tyr
1160 1165 1170
Leu Asn Lys Ile Glu Ser Tyr Leu Ile Asp Ile Asn Trp Lys Leu
1175 1180 1185
Ala Ile Gln Met Ala Arg Phe Glu Arg Asp Met His Tyr Ile Val
1190 1195 1200
Asn Gly Leu Arg Glu Leu Gly Ile Ile Lys Leu Ser Gly Tyr Asn
1205 1210 1215
Thr Gly Ile Ser Arg Ala Tyr Pro Lys Arg Asn Gly Ser Asp Gly
1220 1225 1230
Phe Tyr Thr Thr Thr Ala Tyr Tyr Lys Phe Phe Asp Glu Glu Ser
1235 1240 1245
Tyr Lys Lys Phe Glu Lys Ile Cys Tyr Gly Phe Gly Ile Asp Leu
1250 1255 1260
Ser Glu Asn Ser Glu Ile Asn Lys Pro Glu Asn Glu Ser Ile Arg
1265 1270 1275
Asn Tyr Ile Ser His Phe Tyr Ile Val Arg Asn Pro Phe Ala Asp
1280 1285 1290
Tyr Ser Ile Ala Glu Gln Ile Asp Arg Val Ser Asn Leu Leu Ser
1295 1300 1305
Tyr Ser Thr Arg Tyr Asn Asn Ser Thr Tyr Ala Ser Val Phe Glu
1310 1315 1320
Val Phe Lys Lys Asp Val Asn Leu Asp Tyr Asp Glu Leu Lys Lys
1325 1330 1335
Lys Phe Lys Leu Ile Gly Asn Asn Asp Ile Leu Glu Arg Leu Met
1340 1345 1350
Lys Pro Lys Lys Val Ser Val Leu Glu Leu Glu Ser Tyr Asn Ser
1355 1360 1365
Asp Tyr Ile Lys Asn Leu Ile Ile Glu Leu Leu Thr Lys Ile Glu
1370 1375 1380
Asn Thr Asn Asp Thr Leu
1385
<210> 60
<211> 1300
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 60
Met Ser Ile Tyr Gln Glu Phe Val Asn Lys Tyr Ser Leu Ser Lys Thr
1 5 10 15
Leu Arg Phe Glu Leu Ile Pro Gln Gly Lys Thr Leu Glu Asn Ile Lys
20 25 30
Ala Arg Gly Leu Ile Leu Asp Asp Glu Lys Arg Ala Lys Asp Tyr Lys
35 40 45
Lys Ala Lys Gln Ile Ile Asp Lys Tyr His Gln Phe Phe Ile Glu Glu
50 55 60
Ile Leu Ser Ser Val Cys Ile Ser Glu Asp Leu Leu Gln Asn Tyr Ser
65 70 75 80
Asp Val Tyr Phe Lys Leu Lys Lys Ser Asp Asp Asp Asn Leu Gln Lys
85 90 95
Asp Phe Lys Ser Ala Lys Asp Thr Ile Lys Lys Gln Ile Ser Glu Tyr
100 105 110
Ile Lys Asp Ser Glu Lys Phe Lys Asn Leu Phe Asn Gln Asn Leu Ile
115 120 125
Asp Ala Lys Lys Gly Gln Glu Ser Asp Leu Ile Leu Trp Leu Lys Gln
130 135 140
Ser Lys Asp Asn Gly Ile Glu Leu Phe Lys Ala Asn Ser Asp Ile Thr
145 150 155 160
Asp Ile Asp Glu Ala Leu Glu Ile Ile Lys Ser Phe Lys Gly Trp Thr
165 170 175
Thr Tyr Phe Lys Gly Phe His Glu Asn Arg Lys Asn Val Tyr Ser Ser
180 185 190
Asn Asp Ile Pro Thr Ser Ile Ile Tyr Arg Ile Val Asp Asp Asn Leu
195 200 205
Pro Lys Phe Leu Glu Asn Lys Ala Lys Tyr Glu Ser Leu Lys Asp Lys
210 215 220
Ala Pro Glu Ala Ile Asn Tyr Glu Gln Ile Lys Lys Asp Leu Ala Glu
225 230 235 240
Glu Leu Thr Phe Asp Ile Asp Tyr Lys Thr Ser Glu Val Asn Gln Arg
245 250 255
Val Phe Ser Leu Asp Glu Val Phe Glu Ile Ala Asn Phe Asn Asn Tyr
260 265 270
Leu Asn Gln Ser Gly Ile Thr Lys Phe Asn Thr Ile Ile Gly Gly Lys
275 280 285
Phe Val Asn Gly Glu Asn Thr Lys Arg Lys Gly Ile Asn Glu Tyr Ile
290 295 300
Asn Leu Tyr Ser Gln Gln Ile Asn Asp Lys Thr Leu Lys Lys Tyr Lys
305 310 315 320
Met Ser Val Leu Phe Lys Gln Ile Leu Ser Asp Thr Glu Ser Lys Ser
325 330 335
Phe Val Ile Asp Lys Leu Glu Asp Asp Ser Asp Val Val Thr Thr Met
340 345 350
Gln Ser Phe Tyr Glu Gln Ile Ala Ala Phe Lys Thr Val Glu Glu Lys
355 360 365
Ser Ile Lys Glu Thr Leu Ser Leu Leu Phe Asp Asp Leu Lys Ala Gln
370 375 380
Lys Leu Asp Leu Ser Lys Ile Tyr Phe Lys Asn Asp Lys Ser Leu Thr
385 390 395 400
Asp Leu Ser Gln Gln Val Phe Asp Asp Tyr Ser Val Ile Gly Thr Ala
405 410 415
Val Leu Glu Tyr Ile Thr Gln Gln Ile Ala Pro Lys Asn Leu Asp Asn
420 425 430
Pro Ser Lys Lys Glu Gln Glu Leu Ile Ala Lys Lys Thr Glu Lys Ala
435 440 445
Lys Tyr Leu Ser Leu Glu Thr Ile Lys Leu Ala Leu Glu Glu Phe Asn
450 455 460
Lys His Arg Asp Ile Asp Lys Gln Cys Arg Phe Glu Glu Ile Leu Ala
465 470 475 480
Asn Phe Ala Ala Ile Pro Met Ile Phe Asp Glu Ile Ala Gln Asn Lys
485 490 495
Asp Asn Leu Ala Gln Ile Ser Ile Lys Tyr Gln Asn Gln Gly Lys Lys
500 505 510
Asp Leu Leu Gln Ala Ser Ala Glu Asp Asp Val Lys Ala Ile Lys Asp
515 520 525
Leu Leu Asp Gln Thr Asn Asn Leu Leu His Lys Leu Lys Ile Phe His
530 535 540
Ile Ser Gln Ser Glu Asp Lys Ala Asn Ile Leu Asp Lys Asp Glu His
545 550 555 560
Phe Tyr Leu Val Phe Glu Glu Cys Tyr Phe Glu Leu Ala Asn Ile Val
565 570 575
Pro Leu Tyr Asn Lys Ile Arg Asn Tyr Ile Thr Gln Lys Pro Tyr Ser
580 585 590
Asp Glu Lys Phe Lys Leu Asn Phe Glu Asn Ser Thr Leu Ala Asn Gly
595 600 605
Trp Asp Lys Asn Lys Glu Pro Asp Asn Thr Ala Ile Leu Phe Ile Lys
610 615 620
Asp Asp Lys Tyr Tyr Leu Gly Val Met Asn Lys Lys Asn Asn Lys Ile
625 630 635 640
Phe Asp Asp Lys Ala Ile Lys Glu Asn Lys Gly Glu Gly Tyr Lys Lys
645 650 655
Ile Val Tyr Lys Leu Leu Pro Gly Ala Asn Lys Met Leu Pro Lys Val
660 665 670
Phe Phe Ser Ala Lys Ser Ile Lys Phe Tyr Asn Pro Ser Glu Asp Ile
675 680 685
Leu Arg Ile Arg Asn His Ser Thr His Thr Lys Asn Gly Ser Pro Gln
690 695 700
Lys Gly Tyr Glu Lys Phe Glu Phe Asn Ile Glu Asp Cys Arg Lys Phe
705 710 715 720
Ile Asp Phe Tyr Lys Gln Ser Ile Ser Lys His Pro Glu Trp Lys Asp
725 730 735
Phe Gly Phe Arg Phe Ser Asp Thr Gln Arg Tyr Asn Ser Ile Asp Glu
740 745 750
Phe Tyr Arg Glu Val Glu Asn Gln Gly Tyr Lys Leu Thr Phe Glu Asn
755 760 765
Ile Ser Glu Ser Tyr Ile Asp Ser Val Val Asn Gln Gly Lys Leu Tyr
770 775 780
Leu Phe Gln Ile Tyr Asn Lys Asp Phe Ser Ala Tyr Ser Lys Gly Arg
785 790 795 800
Pro Asn Leu His Thr Leu Tyr Trp Lys Ala Leu Phe Asp Glu Arg Asn
805 810 815
Leu Gln Asp Val Val Tyr Lys Leu Asn Gly Glu Ala Glu Leu Phe Tyr
820 825 830
Arg Lys Gln Ser Ile Pro Lys Lys Ile Thr His Pro Ala Lys Glu Ala
835 840 845
Ile Ala Asn Lys Asn Lys Asp Asn Pro Lys Lys Glu Ser Val Phe Glu
850 855 860
Tyr Asp Leu Ile Lys Asp Lys Arg Phe Thr Glu Asp Lys Phe Phe Phe
865 870 875 880
His Cys Pro Ile Thr Ile Asn Phe Lys Ser Ser Gly Ala Asn Lys Phe
885 890 895
Asn Asp Glu Ile Asn Leu Leu Leu Lys Glu Lys Ala Asn Asp Val His
900 905 910
Ile Leu Ser Ile Asp Arg Gly Glu Arg His Leu Ala Tyr Tyr Thr Leu
915 920 925
Val Asp Gly Lys Gly Asn Ile Ile Lys Gln Asp Thr Phe Asn Ile Ile
930 935 940
Gly Asn Asp Arg Met Lys Thr Asn Tyr His Asp Lys Leu Ala Ala Ile
945 950 955 960
Glu Lys Asp Arg Asp Ser Ala Arg Lys Asp Trp Lys Lys Ile Asn Asn
965 970 975
Ile Lys Glu Met Lys Glu Gly Tyr Leu Ser Gln Val Val His Glu Ile
980 985 990
Ala Lys Leu Val Ile Glu Tyr Asn Ala Ile Val Val Phe Glu Asp Leu
995 1000 1005
Asn Phe Gly Phe Lys Arg Gly Arg Phe Lys Val Glu Lys Gln Val
1010 1015 1020
Tyr Gln Lys Leu Glu Lys Met Leu Ile Glu Lys Leu Asn Tyr Leu
1025 1030 1035
Val Phe Lys Asp Asn Glu Phe Asp Lys Thr Gly Gly Val Leu Arg
1040 1045 1050
Ala Tyr Gln Leu Thr Ala Pro Phe Glu Thr Phe Lys Lys Met Gly
1055 1060 1065
Lys Gln Thr Gly Ile Ile Tyr Tyr Val Pro Ala Gly Phe Thr Ser
1070 1075 1080
Lys Ile Cys Pro Val Thr Gly Phe Val Asn Gln Leu Tyr Pro Lys
1085 1090 1095
Tyr Glu Ser Val Ser Lys Ser Gln Glu Phe Phe Ser Lys Phe Asp
1100 1105 1110
Lys Ile Cys Tyr Asn Leu Asp Lys Gly Tyr Phe Glu Phe Ser Phe
1115 1120 1125
Asp Tyr Lys Asn Phe Gly Asp Lys Ala Ala Lys Gly Lys Trp Thr
1130 1135 1140
Ile Ala Ser Phe Gly Ser Arg Leu Ile Asn Phe Arg Asn Ser Asp
1145 1150 1155
Lys Asn His Asn Trp Asp Thr Arg Glu Val Tyr Pro Thr Lys Glu
1160 1165 1170
Leu Glu Lys Leu Leu Lys Asp Tyr Ser Ile Glu Tyr Gly His Gly
1175 1180 1185
Glu Cys Ile Lys Ala Ala Ile Cys Gly Glu Ser Asp Lys Lys Phe
1190 1195 1200
Phe Ala Lys Leu Thr Ser Val Leu Asn Thr Ile Leu Gln Met Arg
1205 1210 1215
Asn Ser Lys Thr Gly Thr Glu Leu Asp Tyr Leu Ile Ser Pro Val
1220 1225 1230
Ala Asp Val Asn Gly Asn Phe Phe Asp Ser Arg Gln Ala Pro Lys
1235 1240 1245
Asn Met Pro Gln Asp Ala Asp Ala Asn Gly Ala Tyr His Ile Gly
1250 1255 1260
Leu Lys Gly Leu Met Leu Leu Gly Arg Ile Lys Asn Asn Gln Glu
1265 1270 1275
Gly Lys Lys Leu Asn Leu Val Ile Lys Asn Glu Glu Tyr Phe Glu
1280 1285 1290
Phe Val Gln Asn Arg Asn Asn
1295 1300
<210> 61
<211> 7403
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 61
tatccggtcg aatcgagaat gacgaccgct acgtcttgga ctacgaagcc gtggcccttg 60
ccgatgctct cggtgtggat gttgccgacc tgttccgcaa gatcgattgc cccaagaacc 120
tgctgcgcag gcgggcaggg taggggagcg gtttccggcg gagattttcg gaggcgccgg 180
taacgttatg tcggggaatt tgctatacat cgacgataat tagttttgtt gattcaggat 240
cgaaatgcgc tcaaacaaag aacgttccgc gtttccctca tgcgctacta cgcccacacc 300
gccatctttc ggcacgcaaa caaagcagat gggttgcctg tcaatgggtg atcattgcct 360
gaagttacca tccatcaata atataaatca tccttactcc gaatgtccct caatcgcatc 420
tatcaaggcc gcgtggcggc cgtcgaaaca ggaacggcct tagcgaaagg taatgtcgaa 480
tggatgcctg ccgcaggagg cgacgaagtt ctctggcagc accacgaact tttccaagct 540
gccatcaact actatctcgt cgccctgctc gcactcgccg acaaaaacaa tcccgtactt 600
ggcccgctga tcagccagat ggataatccc caaagccctt accatgtctg gggaagtttc 660
cgccgccaag gacgtcagcg cacaggtctc agtcaagccg ttgcacctta tatcacgccg 720
ggcaataacg ctcccaccct tgacgaagtt ttccgctcca ttcttgcggg caacccaacc 780
gaccgcgcaa ctttggacgc tgcactcatg caattgctca aggcttgtga cggcgcgggc 840
gctatccagc aggaaggtcg ttcctactgg cccaaattct gcgatcctga ctccactgcc 900
aacttcgcgg gagatccggc catgctccgg cgtgaacaac accgcctcct ccttccgcaa 960
gttctccacg atccggcgat tactcacgac agtcctgccc ttggctcgtt cgacacttat 1020
tcgattgcta cccccgacac cagaactcct caactcaccg gccccaaggc acgcgcccgt 1080
cttgagcagg cgatcaccct ctggcgcgtc cgtcttcccg aatcggctgc tgacttcgat 1140
cgccttgcca gttccctcaa aaaaattccg gacgacgatt ctcgccttaa ccttcagggc 1200
tacgtcggca gcagtgcgaa aggcgaagtt caggcccgtc ttttcgccct tctgctattc 1260
cgtcacctgg agcgttcctc ctttacgctt ggccttctcc gttccgccac cccgccgccc 1320
aagaacgctg aaacacctcc tcccgccggc gttcctttac ctgcggcgtc cgcagccgat 1380
ccggtgcgga tagcccgtgg caaacgcagt tttgtttttc gcgcattcac cagtctcccc 1440
tgctggcatg gcggtgataa catccatccc acctggaagt cattcgacat cgcagcgttc 1500
aaatatgccc tcacggtcat caaccagatc gaggaaaaga cgaaagaacg ccaaaaagaa 1560
tgtgcggaac ttgaaactga tttcgactac atgcacggac ggctcgccaa gattccggta 1620
aaatacacga ccggcgaagc cgaaccgccc cccattctcg caaacgatct ccgcatcccc 1680
ctcctccgcg aacttctcca gaatatcaag gtcgacaccg cactcaccga tggcgaagcc 1740
gtctcctatg gtctccaacg ccgcaccatt cgcggtttcc gcgagctgcg ccgcatctgg 1800
cgcggccatg cccccgctgg cacggtcttt tccagcgagt tgaaagaaaa actagccggc 1860
gaactccgcc agttccagac cgacaactcc accaccatcg gcagcgtcca actcttcaac 1920
gaactcatcc aaaacccgaa atactggccc atctggcagg ctcctgacgt cgaaaccgcc 1980
cgccaatggg ccgatgccgg ttttgccgac gatccgctcg ccgcccttgt gcaagaagcc 2040
gaactccagg aagacatcga cgccctcaag gctccagtca aactcactcc ggccgatcct 2100
gagtattcaa gaaggcaata cgatttcaat gccgtcagca aattcggggc cggctcccgc 2160
tccgccaatc gccacgaacc cgggcagacg gagcgcggcc acaacacctt taccaccgaa 2220
atcgccgccc gtaacgcggc ggacgggaac cgctggcggg caacccacgt ccgcatccat 2280
tactccgctc cccgccttct tcgtgacgga ctccgccgac ctgacaccga cggcaacgaa 2340
gccctggaag ccgtcccttg gctccagccc atgatggaag ccctcgcccc tctcccgacg 2400
cttccgcaag acctcacagg catgccggtc ttcctcatgc ccgacgtcac cctttccggt 2460
gagcgtcgca tcctcctcaa tcttcctgtc accctcgaac cagccgctct tgtcgaacaa 2520
ctgggcaacg ccggtcgctg gcaaaaccag ttcttcggct cccgcgaaga tccattcgct 2580
ctccgatggc ccgccgacgg tgctgtaaaa accgccaagg ggaaaaccca cataccttgg 2640
caccaggacc gcgatcactt caccgtactc ggcgtggatc tcggcacgcg cgatgccggg 2700
gcgctcgctc ttctcaacgt cactgcgcaa aaaccggcca agccggtcca ccgcatcatt 2760
ggtgaggccg acggacgcac ctggtatgcc agccttgccg acgctcgcat gatccgcctg 2820
cccggggagg atgcccggct ctttgtccgg ggaaaactcg ttcaggaacc ctatggtgaa 2880
cgcgggcgaa acgcgtctct tctcgaatgg gaagacgccc gcaatatcat ccttcgcctt 2940
ggccaaaatc ccgacgaact cctcggcgcc gatccccggc gccattcgta tccggaaata 3000
aacgataaac ttctcgtcgc ccttcgccgc gctcaggccc gtcttgcccg tctccagaac 3060
cggagctggc ggttgcgcga ccttgcagaa tcggacaagg cccttgatga aatccatgcc 3120
gagcgtgccg gggagaagcc ttctccgctt ccgcccttgg ctcgcgacga tgccatcaaa 3180
agcaccgacg aagccctcct ttcccagcgt gacatcatcc ggcgatcctt cgttcagatc 3240
gccaacttga tccttcccct tcgcggacgc cgatgggaat ggcggcccca tgtcgaggtc 3300
ccggattgcc acatccttgc gcagagcgat cccggtacgg atgacaccaa gcgtcttgtc 3360
gccggacaac gcggcatctc tcacgagcgt atcgagcaaa tcgaagaact ccgtcgtcgc 3420
tgccaatccc tcaaccgtgc cctgcgtcac aaacccggag agcgtcccgt gctcggacgc 3480
cccgccaagg gcgaggaaat cgccgatccc tgtcccgcgc tcctcgaaaa gatcaaccgt 3540
ctccgggacc agcgcgttga ccaaaccgcg catgccatcc tcgccgccgc tctcggtgtt 3600
cgactccgcg ccccctcaaa agaccgcgcc gaacgccgcc atcgcgacat ccatggcgaa 3660
tacgaacgct ttcgtgcgcc cgctgatttt gtcgtcatcg aaaacctctc ccgttatctc 3720
agctcgcagg atcgtgctcg tagtgaaaac acccgtctca tgcagtggtg ccatcgccag 3780
atcgtgcaaa aactccgtca gctctgcgag acctacggca tccccgtcct cgccgtcccg 3840
gcggcctact catcgcgttt ttcttcccgg gacggctcgg ccggattccg ggccgtccat 3900
ctgacaccgg accaccgtca ccggatgcca tggagccgca tcctcgcccg cctcaaggcc 3960
cacgaggaag acggaaaaag actcgaaaag acggtgctcg acgaggctcg cgccgtccgg 4020
ggactctttg accggctcga ccggttcaac gccgggcatg tcccgggaaa accttggcgc 4080
acgctcctcg cgccgctccc cggcggccct gtgtttgtcc ccctcgggga cgccacaccc 4140
atgcaggccg atctgaacgc cgccatcaac atcgccctcc ggggcatcgc ggctcccgac 4200
cgccacgaca tccatcaccg gctccgtgcc gaaaacaaaa aacgcatcct gagcttgcgt 4260
ctcggcactc agcgcgagaa agcccgctgg cctggaggag ctccggcggt gacactctcc 4320
actccgaaca acggcgcctc tcccgaagat tccgatgcgt tgcccgaacg ggtatccaac 4380
ctgtttgtgg acatcgccgg tgtcgccaac ttcgagcgag tcacgatcga aggagtctcg 4440
caaaaattcg ccaccgggcg tggcctttgg gcctccgtca agcaacgtgc atggaaccgc 4500
gttgccagac tcaacgagac agtaacagat aacaacagga acgaagagga ggacgacatt 4560
ccgatgtaac cattgcttca ttacatctga gtctcccctc aatccctctg ccccatgcgt 4620
gatataacct ccacctcatg tcccggatcg gcgccggcaa cctgtagttc ccttccatcc 4680
tccaacactc ccgcagatcg cgatccgctg ccgccgatgc cggtgcgccg ccttcacaac 4740
tatctctact gtccgcggct tttttatctc cagtgggtcg agaatctctt tgaggaaaat 4800
gccgacacca ttgccggcag cgccgtgcat cgtcacgccg acaaacctac gcgttacgat 4860
gatgaaaaag ccgaggcact tcgcactggt ctccctgaag gcgcgcacat acgcagcctt 4920
cgcctggaaa acgcccaact cggtctcgtt ggcgtggtgg atatcgtgga gggaggcccc 4980
gacggactcg aactcgtcga ctacaaaaaa ggttccgcct tccgcctcga cgacggcacg 5040
ctcgctccca aggaaaacga caccgtgcaa cttgccgcct acgctcttct cctggctgcc 5100
gatggtgcgc gcgttgcgcc catggcgacg gtctattacg ctgccgatcg ccggcgtgtc 5160
accttcccgc tcgatgacgc cctctacgcc cgcacccgtt ccgccctcga agaggcccgc 5220
gccgttgcaa cctcggggcg catacctccg ccgctcgtct ctgacgtccg ctgcctccat 5280
tgttcctcct atgcgctttg ccttccccgc gagtccgcct ggtggtgccg ccatcgcagc 5340
acgccgcggg gagccggcca cacccccatg ttgccgggct ttgaggatga cgccgccgcc 5400
attcaccaaa tctccgaacc tgacaccgag ccaccacccg atcttgccag ccagcctccc 5460
cgtcccccgc ggctcgatgg agaattgttg gttgtccaga ctccgggagc gatgatcgga 5520
caaagcggcg gtgagtttac cgtgtccgtc aagggtgagg ttttgcgcaa gcttccggtt 5580
catcaactcc gggccattta cgtttacgga gccgtgcaac tcacggcgca tgctgtgcag 5640
accgcccttg aggaggatat cgacgtctcc tattttgcgc ccagcggccg ctttcttggc 5700
ctcctccgcg gcctgcccgc atccggcgtg gatgcgcgtc tcgggcaata caccctgttt 5760
cgcgaaccct ttggccgtct ccgtctcgcc tgcgaggcga ttcgggccaa gatccataac 5820
cagcgcgtcc tcctcatgcg taacggcgag cccggggagg gcgtcttgcg cgaactcgcc 5880
cgtctgcgcg acgccaccag tgaggcgact tcgctcgacg aactcctcgg catcgagggc 5940
atcgccgcgc atttctattt ccagtatttt cccaccatgc tgaaagaacg ggcggcctgg 6000
gcctttgatt tttccggacg caatcgccgc ccgccgcgcg acccggtcaa cgccctgctt 6060
tcgttcggtt acagcgtgtt gtccaaggaa cttgccggcg tctgccacgc tgttggccta 6120
gacccgtttt tcggcttcat gcaccagccg cgttacgggc gccccgcact cgctctcgat 6180
ctgatggagg agtttcgccc tctcatcgcc gacagtgttg ccctgaatct catcaaccgt 6240
ggcgaactcg acgaagggga ctttatccgg tcggccaatg gcaccgcgct caatgatcgg 6300
ggccgccggc gtttttggga ggcatggttc cggcgtctcg acagcgaagt cagccatcct 6360
gaatttggtt acaagatgag ctatcgacgg atgcttgaag tgcaggcgcg ccagctatgg 6420
cgctatgtgc gcggtgacgc cttccgctac cacggattca ccacccgttg attccgatgt 6480
cagatccccg ccgccgttat cttgtgtgtt acgacatcgc caatccgaag cgattgcgcc 6540
aagtggccaa gctgctggag agctatggca cgcgtctgca atactcggtt ttcgaatgtc 6600
ctttggacga tcttcgtctt gaacaggcga aggctgattt gcgcgacacg attaatgccg 6660
accaagacca ggtgttattt gtttcgcttg gccccgaagc caacgatgcc acgttgatca 6720
tcgccacgct tgggctccct tataccgtgc gctcgcgagt gacgattatc tgacccataa 6780
cccacgtgtt gaagaggctg aaaacagacg gacctctatg aagaacaatt gacgttttgg 6840
ccgaactcag cagaccttta tgcggctaag gccaatgatc atccatccta ccgccattgg 6900
gctggagacg ttttttgaaa cggcgagtgc tgcggatagc gagtttctct tggggaggcg 6960
ctcgcggcca cttttacaga ggagatgttc gggcgaactg gccgacctaa caaggcgtac 7020
ccggctcaaa atcgaggcac gctcgcacgg gatgatgtaa ttcgttgttt ttcagcatac 7080
cgtgcgagca cgggccgcag cgaatgccgt ttcacgaatc gtcaggcggc ggggagaagt 7140
catttaataa ggccactgtt aaaagccgca gcgaatgccg tttcacgaat cgtcaggcgg 7200
gcagtggatg tttttccatg aggcgaagaa tttcatcgcc gcagtgaatg ccgtttcacc 7260
attgatgaag aatgcgaggt gaaaacagag aaattgggtc aactctatca ctcttattca 7320
gccatcgttt caagaaagga tacctcgtat tggatacaac acagctcgtt cgttctctct 7380
acctccctcg acaatctcaa gga 7403
<210> 62
<211> 6789
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 62
taataaaatt gaaatatcac tatggattat tgtaatatta ccataaagat aggtgacgtt 60
tttttgaaaa ttgtaaacct aatttgaaga aaaccaatta aaaatcgctt cggctttttt 120
ttaagtgcca ggtagcattg atgctaaccc atgtgtaata aaggtttgtt ttccttcggg 180
gcacgaacac attataaggg aaacctaaag attccctttc ttgtttaata ttataaccag 240
tgaaaataag aataatgcac ctaaaactaa tatacagaaa ataagaatta aaagtactaa 300
tatatacatc atatgttatc ctccaatgct ttatttttta ataattgatg ttagtattag 360
ttttatttta atttctaaac ataagaattt gaaaaggatg tgtttattat ggcgacacgc 420
agttttattt taaaaattga accaaatgaa gaagttaaaa agggattatg gaagacgcat 480
gaggtattga atcatggaat tgcctactac atgaatattc tgaaactaat tagacaggaa 540
gctatttatg aacatcatga acaagatcct aaaaatccga aaaaagtttc aaaagcagaa 600
atacaagccg agttatggga ttttgtttta aaaatgcaaa aatgtaatag ttttacacat 660
gaagttgaca aagatgttgt ttttaacatc ctgcgtgaac tatatgaaga gttggtccct 720
agttcagtcg agaaaaaggg tgaagccaat caattatcga ataagtttct gtacccgcta 780
gttgatccga acagtcaaag tgggaaaggg acggcatcat ccggacgtaa acctcggtgg 840
tataatttaa aaatagcagg cgacccatcg tgggaggaag aaaagaaaaa atgggaagag 900
gataaaaaga aagatcccct tgctaaaatc ttaggtaagt tagcagaata tgggcttatt 960
ccgctattta ttccatttac tgacagcaac gaaccaattg taaaagaaat taaatggatg 1020
gaaaaaagtc gtaatcaaag tgtccggcga cttgataagg atatgtttat ccaagcatta 1080
gagcgttttc tttcatggga aagctggaac cttaaagtaa aggaagagta tgaaaaagtt 1140
gaaaaggaac acaaaacact agaggaaagg ataaaagagg acattcaagc atttaaatcc 1200
cttgaacaat atgaaaaaga acggcaggag caacttctta gagatacatt gaatacaaat 1260
gaataccgat taagcaaaag aggattacgt ggttggcgtg aaattatcca aaaatggcta 1320
aagatggatg aaaatgaacc atcagaaaaa tatttagaag tatttaaaga ttatcaacgg 1380
aaacatccac gagaagccgg ggactattct gtctatgaat ttttaagcaa gaaagaaaat 1440
cattttattt ggcgaaatca tcctgaatat ccttatttgt atgctacatt ttgtgaaatt 1500
gacaaaaaaa agaaagacgc taagcaacag gcaactttta ctttggctga cccgattaac 1560
catccgttat gggtacgatt tgaagaaaga agcggttcga acttaaacaa atatcgaatt 1620
ttaacagagc aattacacac tgaaaagtta aaaaagaaat taacagttca acttgatcgt 1680
ttaatttatc caactgaatc cggcggttgg gaggaaaaag gtaaagtaga tatcgttttg 1740
ttgccgtcaa gacaatttta taatcaaatc ttccttgata tagaagaaaa ggggaaacat 1800
gcttttactt ataaggatga aagtattaaa ttccccctta aaggtacact tggtggtgca 1860
agagtgcagt ttgaccgtga ccatttgcgg agatatccgc ataaagtaga atcaggaaat 1920
gttggacgga tttattttaa catgacagta aatattgaac caactgagag ccctgttagt 1980
aagtctttga aaatacatag ggacgatttc cccaagttcg ttaattttaa accgaaagag 2040
ctcaccgaat ggataaaaga tagtaaaggg aaaaaattaa aaagtggtat agaatccctt 2100
gaaattggtc tacgggtgat gagtatcgac ttaggtcaac gtcaagcggc tgctgcatcg 2160
atttttgaag tagttgatca gaaaccggat attgaaggga agttattttt tccaatcaaa 2220
ggaactgagc tttatgctgt tcaccgggca agttttaaca ttaaattacc gggtgaaaca 2280
ttagtaaaat cacgggaagt attgcggaaa gctcgggagg acaacttaaa attaatgaat 2340
caaaagttaa actttctaag aaatgttcta catttccaac agtttgaaga tatcacagaa 2400
agagagaagc gtgtaactaa atggatttct agacaagaaa atagtgatgt tcctcttgta 2460
tatcaagatg agctaattca aattcgtgaa ttaatgtata aaccctataa agattgggtt 2520
gcctttttaa aacaactcca taaacggcta gaagtcgaga ttggcaaaga ggttaagcat 2580
tggcgaaaat cattaagtga cgggagaaaa ggtctttacg gaatctccct aaaaaatatt 2640
gatgaaattg atcgaacaag gaaattcctt ttaagatgga gcttacgtcc aacagaacct 2700
ggggaagtaa gacgcttgga accaggacag cgttttgcga ttgatcaatt aaaccaccta 2760
aatgcattaa aagaagatcg attaaaaaag atggcaaata cgattatcat gcatgcctta 2820
ggttactgtt atgatgtaag aaagaaaaag tggcaggcaa aaaatccagc atgtcaaatt 2880
attttatttg aagatttatc taactacaat ccttacgagg aaaggtcccg ttttgaaaac 2940
tcaaaactga tgaagtggtc acggagagaa attccacgac aagtcgcctt acaaggtgaa 3000
atttacggat tacaagttgg ggaagtaggt gcccaattca gttcaagatt ccatgcgaaa 3060
accgggtcgc cgggaattcg ttgcagtgtt gtaacgaaag aaaaattgca ggataatcgc 3120
ttttttaaaa atttacaaag agaaggacga cttactcttg ataaaatcgc agttttaaaa 3180
gaaggagact tatatccaga taaaggtgga gaaaagttta tttctttatc aaaggatcga 3240
aagttggtaa ctacgcatgc tgatattaac gcggcccaaa atttacagaa gcgtttttgg 3300
acaagaacac atggatttta taaagtttac tgcaaagcct atcaggttga tggacaaact 3360
gtttatattc cggagagcaa ggaccaaaaa caaaaaataa ttgaagaatt tggggaaggc 3420
tattttattt taaaagatgg tgtatatgaa tggggtaatg cggggaaact aaaaattaaa 3480
aaaggttcct ctaaacaatc atcgagtgaa ttagtagatt cggacatact gaaagattca 3540
tttgatttag caagtgaact taagggagag aaactcatgt tatatcgaga tccgagtgga 3600
aacgtatttc cttccgacaa gtggatggca gcaggagtat tttttggcaa attagaaaga 3660
atattgattt ctaagttaac aaatcaatac tcaatatcaa caatagaaga tgattcttca 3720
aaacaatcaa tgtaaaagtt tgcccgtata agaacttaat taattaggat ggtaggatgt 3780
tactaaatat gtctgtaggc atcattccta ctatccgttt tgtccgaata tcagagcatt 3840
aggtgaggaa tggtaagaaa ggaaaattta tatgaaccaa ccgattccta ttcgaatgtt 3900
aaatgaaata caatattgtg agcgactttt ttactttatg catgtccaaa agctatttga 3960
tgagaatgca gatacagttg aaggaagtgc acagcatgag cgggcagaaa gaagcaaaag 4020
accaagtaaa atgggaccaa aggaattatg gggtgaggcg ccaagaagtc ttaagcttgg 4080
tgatgagctg ttaaatatta ccggtgttct tgatgccata agtcatgaag agaacagttg 4140
gatcccggtt gaatcaaaac acagttccgc accggatgga ttgaaccctt ttaaagtaga 4200
tggctttcta cttgacgggt ctgcatggcc aaacgatcaa attcaacttt gtgcacaagg 4260
cttgctcttg aatgccaatg gatacccgtg tgattatggg tatttatttt atcgtggtaa 4320
taagaaaaag gtgaaaattt attttactga agatttaatc gctgccacaa agtactatat 4380
taaaaaagca cacgagatac tagtattatc tggtgatgaa tcagctattc ctaagccttt 4440
aattgattct aataagtgtt ttcgctgttc tttaaactat atctgtcttc cggatgaaac 4500
gaactatcta ttaggggcaa gttcaacaat tcgtaaaatt gtgccttcaa ggacagatgg 4560
tggcgtttta tatgtatcag agtctggtac aaaattagga aaatcgggtg aggagttaat 4620
cattcagtat aaagatggcc aaaagcaggg tgttcctata aaagatatta ttcaagtttc 4680
gttaattgga aatgttcaat gctcaacgca attacttcat tttttaatgc aatcaaatat 4740
tcctgtaagt tatttatcat cccacggtcg tttgattggt gtcagttcat ctttagttac 4800
aaaaaatgtt ttaacaaggc agcaacagtt cattaaattt acaaatcctg agtttggact 4860
aaatctagca aaacaaattg tttatgccaa gattcgaaat caacgaactt tacttagaag 4920
aaatgggggg agtgaggtaa aggagatttt aacagattta aaatctttaa gtgacagtgc 4980
actgaacgca atatcaatag aacaattacg gggtattgaa gggatttctg caaaacatta 5040
tttcgcagga tttccgttta tgttgaaaaa tgaattacgt gaattgaatt taatgaaagg 5100
gcgtaatagg agaccgccaa aagatcctgt aaatgtactt ctttctcttg gttatacttt 5160
attgacacgt gatattcatg ctgcgtgtgg ttcagtcgga ttggatccga tgtttggttg 5220
ttaccatcgt ccagaagcag gtcgaccggc tctagtatta gatgttatgg aaacatttcg 5280
accacttatt gtagacagta ttgtcatccg agctttgaat acgggtgaaa tctcattaaa 5340
agatttttat ataggaaaag atagttgtca attattaaaa catggccgcg attccttttt 5400
tgccatttat gaaagaagaa tgcatgaaac tattaccgat ccaattttcg gctataagat 5460
tagctatcgc cgtatgctcg atttgcacat tcgaatgctt gcaaggttta ttgaagggga 5520
actgccggaa tataaaccat taatgacccg gtgagtttgt ttattaggtt aaaagaaggt 5580
gaagacatgc agcaatacgt ccttgtttct tatgatattt cggaccaaaa aagatggaga 5640
aaagtattta aactgatgaa aggatacgga gaacatgttc aatattccgt attcatatgc 5700
cagttaactg aattacagaa ggcaaaatta caagcctctt tagaagacat tatccatcat 5760
aagaatgacc aagtaatgtt tgttcacatc gggccagtga aagatggtca actatctaaa 5820
aaaatctcaa caattgggaa agaatttgtt ccattggatt taaagcggct tatattttga 5880
aaagatatag caaagaaatc ttatgaaaaa aatacaaaaa tatattgtta aaaaataggg 5940
aatattatat aatggactta cgaggttctg tcttttggtc aggacaaccg tctagctata 6000
agtgctgcag gggtgtgaga aactcctatt gctggacgat gtctctttta tttctttttt 6060
cttggatctg agtacgagca cccacattgg acatttcgca tggtgggtgc tcgtactata 6120
ggtaaaacaa acctttttaa gaagaataca aaaataacca caatattttt taaaaggaat 6180
tttgatggat ttacataacc tctcgcaaca tgcttctaaa acccaagccc accatagccc 6240
aaaaccccct gcggtccaag aaaaaagaaa tgatacgagg cattagcacc ggggagaagt 6300
catttaataa ggccactgtt aaaagtccaa gaaaaaagaa atgatacgag gcattagcac 6360
aacaatataa acgactactt taccgtgttc aagaaaaaag aaatgatatg aggcattagc 6420
acgatgggat gggagagaga ggacagttct actcttgctg tatccagctt cttttacttt 6480
atccggtatc atttcttcac ttctttctgc acataaaaaa gcacctaact atttggataa 6540
gttaagtgct tttatttccg tttgaagttg tctattgctt ttttcttcat atcttcaaat 6600
tttttctgtt tctcagagtc aactttacca actgtaatcc cttttctttt tggcattggg 6660
gtatctttcc accttagtgt gttcataagg cttatattta tcactcattg tattcctcca 6720
acacaattat aatttttccg tcatcctcaa tccaaccgtc aactgtgaca aaagacgaat 6780
ctctcttat 6789
<210> 63
<211> 6214
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 63
gtttcatttg gaaagggaga gcattggctt ttctctttgt aaataaagtg caagctttgt 60
aataagcttc tagtggagaa gtgattgttt gaatcaccca atgcacacgc actaaagtta 120
gacgaaccta taattcgtat tagtaagtat agtacatgaa gaaaaatgca acaagcattt 180
actctctttt aaataaagaa ttgatagctg ttaatattga tagtatatta taccttatag 240
atgttcgatt ttttttgaaa ttcaaaaatc atacttagta aagaaaggaa ataacgtcat 300
ggacaagcga aagcgtagaa gttacgagtt taggtgggaa gcgggaggca ccagtcatgg 360
caatccgtag cataaaacta aaactaaaaa cccacacagg cccggaagcg caaaacctcc 420
gaaaaggaat atggcggacg catcggttgt taaatgaagg cgtcgcctat tacatgaaaa 480
tgctcctgct ctttcgtcag gaaagcactg gtgaacggcc aaaagaagaa ctacaggaag 540
aactgatttg tcacatacgc gaacagcaac aacgaaatca ggcagataaa aatacgcaag 600
cgcttccgct agataaggca ctggaagctt tgcgccaact atatgaactg cttgtcccct 660
cctcggtcgg acaaagtggc gacgcccaga tcatcagccg aaagtttctc agcccgctcg 720
tcgatccgaa cagcgaaggc ggcaaaggta cttcgaaggc aggggcaaaa cccacttggc 780
agaagaaaaa agaagcgaac gacccaacct gggaacagga ttacgaaaaa tggaaaaaaa 840
gacgcgagga agacccaacc gcttctgtga ttactacttt ggaggaatac ggcattagac 900
cgatctttcc cctgtacacg aacaccgtaa cagatatcgc gtggttgcca cttcaatcca 960
atcagtttgt gcgaacctgg gacagagaca tgcttcaaca agcgattgaa agactgctca 1020
gttgggagag ctggaacaaa cgtgtccagg aagagtatgc caagctgaaa gaaaaaatgg 1080
ctcaactgaa cgagcaactc gaaggcggtc aggaatggat cagcttgcta gagcagtacg 1140
aagaaaaccg agagcgagag cttagggaaa acatgaccgc tgccaatgac aagtatcgga 1200
ttaccaagcg gcaaatgaaa ggctggaacg agctgtacga gctatggtca acctttcccg 1260
ccagtgccag tcacgagcaa tacaaagagg cgctcaagcg tgtgcagcag cgactgagag 1320
ggcggtttgg ggatgctcat ttcttccagt atctgatgga agagaagaac cgcctgatct 1380
ggaaggggaa tccgcagcgt atccattatt ttgtcgcgcg caacgaactg acgaaacggc 1440
tggaggaagc caagcaaagc gccacgatga cgttgcccaa tgccaggaag catccattgt 1500
gggtgcgctt cgatgcacgg ggaggaaatt tgcaagacta ctacttgacg gctgaagcgg 1560
acaaaccgag aagcagacgt tttgtaacgt ttagtcagtt gatatggcca agcgaatcgg 1620
gatggatgga aaagaaagac gtcgaggtcg agctagcttt gtccaggcag ttttaccagc 1680
aggtgaagtt gctgaaaaat gacaaaggca agcagaaaat cgagttcaag gataaaggtt 1740
cgggctcgac gtttaacgga cacttggggg gagcaaagct acaactggag cggggcgatt 1800
tggagaagga agaaaaaaac ttcgaggacg gggaaatcgg cagcgtttac cttaacgttg 1860
tcattgattt cgaacctttg caagaagtga aaaatggccg cgtgcaggcg ccgtatggac 1920
aagtactgca actcattcgt cgccccaacg agtttcccaa ggtcactacc tataagtcgg 1980
agcaacttgt tgaatggata aaagcttcgc cacaacactc ggctggggtg gagtcgctgg 2040
catccggttt tcgtgtaatg agcatagacc ttgggctgcg cgcggctgca gcgacttcta 2100
ttttttctgt agaagagagt agcgataaaa atgcggctga tttttcctac tggattgaag 2160
gaacgccgct ggtcgctgtc catcagcgga gctatatgct caggttgcct ggtgaacagg 2220
tagaaaaaca ggtgatggaa aaacgggacg agcggttcca gctacaccaa cgtgtgaagt 2280
ttcaaatcag agtgctcgcc caaatcatgc gtatggcaaa taagcagtat ggagatcgct 2340
gggatgaact cgacagcctg aaacaagcgg ttgagcagaa aaagtcgccg ctcgatcaaa 2400
cagaccggac attttgggag gggattgtct gcgacttaac aaaggttttg cctcgaaacg 2460
aagcggactg ggaacaagcg gtagtgcaaa tacaccgaaa agcagaggaa tacgtcggaa 2520
aagccgttca ggcatggcgc aagcgctttg ctgctgacga gcgaaaaggc atcgcaggtc 2580
tgagcatgtg gaacatagaa gaattggagg gcttgcgcaa gctgttgatt tcctggagcc 2640
gcaggacgag gaatccgcag gaggttaatc gctttgagcg aggccatacc agccaccagc 2700
gtctgttgac ccatatccaa aacgtcaaag aggatcgcct gaagcagtta agtcacgcca 2760
ttgtcatgac tgccttgggg tatgtttacg acgagcggaa acaagagtgg tgcgccgaat 2820
acccggcttg ccaggtcatt ctgtttgaaa atctgagcca gtaccgttct aacctggatc 2880
gctcgaccaa agaaaactcc accttgatga agtgggcgca tcgcagcatt ccgaaatacg 2940
tccacatgca ggcggagcca tacgggattc agattggcga tgtccgggcg gaatattcct 3000
ctcgttttta cgccaagaca ggaacgccag gcattcgttg taaaaaggtg agaggccaag 3060
acctgcaggg cagacggttt gagaacttgc agaagaggtt agtcaacgag caatttttga 3120
cggaagaaca agtgaaacag ctaaggcccg gcgacattgt cccggatgat agcggagaac 3180
tgttcatgac cttgacagac ggaagcggaa gcaaggaggt cgtgtttctc caggccgata 3240
ttaacgcggc gcacaatctg caaaaacgtt tttggcagcg atacaatgaa ctgttcaagg 3300
ttagctgccg cgtcatcgtc cgagacgagg aagagtatct cgttcccaag acaaaatcgg 3360
tgcaggcaaa gctgggcaaa gggctttttg tgaaaaaatc ggatacagcc tggaaagatg 3420
tatatgtgtg ggacagccag gcaaagctta aaggtaaaac aacctttaca gaagagtctg 3480
agtcgcccga acaactggaa gactttcagg agatcatcga ggaagcagaa gaggcgaaag 3540
gaacataccg tacactgttc cgcgatccta gcggagtctt ttttcccgaa tccgtatggt 3600
atccccaaaa agatttttgg ggcgaggtga aaaggaagct gtacggaaaa ttgcgggaac 3660
ggtttttgac aaaggctcgg taagggtgtg caaggagagt gaatggcttg tcctggatac 3720
ctgtccgcat gctaaatgaa attcagtatt gtgagcgact gtaccatatt atgcatgtgc 3780
aggggctgtt tgaggaaagc gcagacacgg tcgaaggagc agcacaacac aagcgtgcag 3840
agacacatct gcgcaaaagc aaggcagcgc cggaagagat gtggggggac gctccgttta 3900
gcttgcagct cggcgaccct gtgcttggca ttacgggaaa gctggatgcc gtctgtctgg 3960
aagaaggtaa gcagtggatt ccggtagaag gaaagcattc ggcgtcgcca gaaggcgggc 4020
agatgttcac tgtaggcgtg tattcgctgg acggttctgc ctggcccaac gaccaaatcc 4080
aattgtgtgc gcaaggcttg ctgcttcgcg cgaatggata tgaatccgat tatggctact 4140
tatactaccg tggcaataaa aagaaggttc gcattccttt ttcgcaggaa ctcatagcgg 4200
ctactcacgc ctgcattcaa aaagctcatc agcttcggga agccgaaatt ccccctccgt 4260
tgcaggagtc gaaaaagtgc tttcgatgct cgttaaatta cgtatgcatg cctgacgaga 4320
cgaattacat gttggggttg agcgcaaaca tcagaaagat tgtgcccagt cgtccagatg 4380
gcggggtact gtatgttaca gagcaggggg caaaactggg cagaagcgga gaaagcttga 4440
ccatcacctg ccggggcgaa aagatagacg aaatcccgat caaagacttg attcacgtga 4500
gcttgatggg gcatgtgcaa tgctctacgc agcttctgca caccttgatg aactgtggcg 4560
tccacgtcag ctacttgact acgcatggca cattgacagg aataatgact ccccctttat 4620
cgaaaaacat tcgaacaaga gccaagcagt ttatcaaatt tcagcacgcg gagatcgccc 4680
ttggaatcgc gagaagggtc gtgtatgcga aaatttccaa tcagcgcacg atgctgcgcc 4740
gcaatggctc accagataaa gcagttttaa aagagttaaa agagcttaga gatcgcgcgt 4800
gggaggcgcc atcactggaa atagtgagag gtatcgaggg acgtgcagca cagttgtaca 4860
tgcagttttt ccctaccatg ttaaagcacc cagtagtaga cggtatggcg atcatgaacg 4920
gtcgcaaccg tcgcccgccc aaagatccgg tcaatgcgct gctctccctc ggctatacgc 4980
ttctttcacg ggatgtttac tccgcatgtg ccaatgtcgg actcgatcca ctgttcggct 5040
ttttccatac gatggagccg ggcagaccag ctttggcact cgatctgatg gaaccgttcc 5100
gcgccttgat tgccgatagc gtagcgatac gtaccttgaa tacggaggaa ctcaccctcg 5160
gggactttta ttggggaaaa gacagttgtt atttgaaaaa ggcaggaaga caaacgtatt 5220
tcgctgccta tgaaagacgg atgaacgaga cgctgacgca tccgcaattt gggtataagc 5280
tcagctatcg ccgtatgctg gagctggaag caaggttttt ggcccggtat ctggatggag 5340
agctggtgga atatacgccg ctcatgacaa ggtaggaaat gaccatgcga caatttgttc 5400
tggtaagcta tgatattgcc gatcaaaaac gttggagaaa agtattcaag ctgatgaagg 5460
ggcaaggcga gcacgtccag tactcggtgt ttctgtgcca actcaccgag attcagcaag 5520
ccaagctaaa ggtaagcctg gcggagctgg ttcaccatgg agaagaccag gtcatgtttg 5580
taaaaatcgg cccagtgacg agagatcaac tggacaagcg gatatctact gttggcaggg 5640
agtttctgcc tcgcgatttg accaaattta tctattaagg aatgaagaaa gctagttgta 5700
acaaaagtgg aaaaagagta aaataaaggt gtcagtcgca cgctataggc cataagtcga 5760
cttacatatc cgtgcgtgtg cattatgggc ccatccacag gtctattccc acggataatc 5820
acgactttcc actaagcttt cgaattttat gatgcgagca tcctctcagg tcaaaaaagc 5880
cgggggatgc tcgaactctt tgtgggcgta ggctttccag agttttttag gggaagaggc 5940
agccgatgga taagaggaat ggcgattgaa ttttggcttg ctcgaaaaac gggtctgtaa 6000
ggcttgcggc tgtaggggtt gagtgggaag gagttcgaaa gcttagtgga aagcttcgtg 6060
gttagcaccg gggagaagtc atttaataag gccactgtta aaagttcgaa agcttagtgg 6120
aaagcttcgt ggttagcacg ctaaagtccg tctaaactac tgagatctta aatcggcgct 6180
caaataaaaa acctcgctaa tgcgaggttt cagc 6214
<210> 64
<211> 12338
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 64
gaagttatgt tgataaaatg gtttatgaaa acgtgagtct gtggtagtat tataaacaat 60
gatggaataa agtgtttttt gcgccgcacg gcatgaattc aggggttagc ttggttttgt 120
gtataaataa atgttctaca tatttatttt gttttttgcg ccgcaaaatg caactgaaag 180
ccgcatctag agcaccctgt agaagacagg gttttgagaa tagcccgaca tagagggcaa 240
tagacacggg gagaagtcat ttaataaggc cactgttaaa agttttgaga atagcccgac 300
atagagggca atagactttt gcttcgtcac ggatggactt cacaatggca acaacgtttt 360
gagaatagcc cgacatagtt atagagatgt ataaatataa ccgataaaca ttgactaatt 420
tgttgaagtc agtgtttatc ggttttttgt gtaaatatag gagttgttag aatgatactt 480
tttgcctaat tttggaactt tatgaggata taagatagac ttgataaaaa ggtaaaagaa 540
aggttaaaga gcatggcagg aatagtgacc tgtgatgaag atgatggtag aattaaaagt 600
gttcttaaag aaaaacaata ttggataagg aaaataattc aatagataaa aaatttaggg 660
ggaaaaatga aaatatcaaa agtcgatcat accagaatgg cggttgctaa aggtaatcaa 720
cacaggagag atgagattag tgggattctc tataaggatc cgacaaagac aggaagtata 780
gattttgatg aacgattcaa aaaactgaat tgttcggcga agatacttta tcatgtattc 840
aatggaattg ctgagggaag caataaatac aaaaatattg ttgataaagt aaataacaat 900
ttagataggg tcttatttac aggtaagagc tatgatcgaa aatctatcat agacatagat 960
actgttctta gaaatgttga gaaaattaat gcatttgatc gaatttcaac agaggaaaga 1020
gaacaaataa ttgacgattt gttagaaata caattgagga aggggttaag gaaaggaaaa 1080
gctggattaa gagaggtatt actaattggt gctggtgtaa tagttagaac cgataagaag 1140
caggaaatag ctgattttct ggagatttta gatgaagatt tcaataagac gaatcaggct 1200
aagaacataa aattgtctat tgagaatcag gggttggtgg tctcgcctgt atcaagggga 1260
gaggaacgga tttttgatgt cagtggcgca caaaagggaa aaagcagcaa aaaagcgcag 1320
gagaaagagg cactatctgc atttctgtta gattatgctg atcttgataa gaatgtcagg 1380
tttgagtatt tacgtaaaat tagaagactg ataaatctat atttctatgt caaaaatgat 1440
gatgttatgt ctttaactga aattccggca gaagtgaatc tggaaaaaga ttttgatatc 1500
tggagagatc acgaacaaag aaaggaagag aatggagatt ttgttggatg tccggacata 1560
cttttggcag atcgtgatgt gaagaaaagt aacagtaagc aggtaaaaat tgcagagagg 1620
caattaaggg agtcaatacg tgaaaaaaat ataaaacgat atagatttag cataaaaacg 1680
attgaaaagg atgatggaac atactttttt gcaaataagc agataagtgt attttggatt 1740
catcgcattg aaaatgctgt agaacgtata ttaggatcta ttaatgataa aaaactgtat 1800
agattacgtt taggatatct aggagaaaaa gtatggaagg acatactcaa ttttctcagc 1860
ataaaataca ttgcagtagg caaggcagta ttcaattttg caatggatga tctgcaggag 1920
aaggatagag atatagaacc cggcaagata tcagaaaatg cagtaaatgg attgacttcg 1980
tttgattatg agcaaataaa ggcagatgag atgctgcaga gagaagttgc tgttaatgta 2040
gcattcgcag caaataatct tgctagagta actgtagata ttccgcaaaa tggagaaaaa 2100
gaggatatcc ttctttggaa taaaagtgac ataaaaaaat acaaaaagaa ttcaaagaaa 2160
ggtattctga aatctatact tcagtttttt ggtggtgctt caacttggaa tatgaaaatg 2220
tttgagattg catatcatga tcagccaggt gattacgaag aaaactacct atatgacatt 2280
attcagatca tttactcgct cagaaataag agctttcatt tcaagacata tgatcatggg 2340
gataagaatt ggaatagaga actgatagga aagatgattg agcatgatgc tgaaagagtc 2400
atttctgttg agagggaaaa gtttcattcc aataacctgc cgatgtttta taaagacgct 2460
gatctaaaga aaatattgga tctcttgtat agcgattatg caggacgtgc atctcaggtt 2520
ccggcattta acactgtctt ggttcgaaag aactttccgg aatttcttag gaaagatatg 2580
ggctacaagg ttcattttaa caatcctgaa gtagagaatc agtggcacag tgcggtgtat 2640
tacctatata aagagattta ttacaatcta tttttgagag ataaagaggt aaagaatctt 2700
ttttatactt cattaaaaaa tataagaagt gaagtttcgg acaaaaaaca aaagttagct 2760
tcagatgatt ttgcatccag gtgtgaagaa atagaggata gaagtcttcc ggaaatttgt 2820
cagataataa tgacagaata caatgcgcag aactttggta atagaaaagt taaatctcag 2880
cgtgttattg aaaaaaataa ggatattttc agacattata aaatgctttt gataaagact 2940
ttagcaggtg ctttttctct ttatttgaag caggaaagat ttgcatttat tggtaaggca 3000
acacctatac catacgaaac aaccgatgtt aagaattttt tgcctgaatg gaaatccgga 3060
atgtatgcat cgtttgtaga ggagataaag aataatcttg atcttcaaga atggtatatc 3120
gtcggacgat tccttaatgg gaggatgctc aatcaattgg caggaagcct gcggtcatac 3180
atacagtatg cggaagatat agaacgtcgt gctgcagaaa ataggaataa gcttttctcc 3240
aagcctgatg aaaagattga agcatgtaaa aaagcggtca gagtgcttga tttgtgtata 3300
aaaatttcaa ctagaatatc tgcggaattt actgactatt ttgatagtga agatgattat 3360
gcagattatc ttgaaaaata tctcaagtat caggatgatg ccattaagga attgtcagga 3420
tcttcgtatg ctgcgttgga tcatttttgc aacaaggatg atctgaaatt tgatatctat 3480
gtaaatgccg gacagaagcc tatcttacag agaaatatcg tgatggcaaa gctttttgga 3540
ccagataaca ttttgtctga agttatggaa aaggtaacag aaagtgccat acgagaatac 3600
tatgactatc tgaagaaagt ttcaggatat cgggtaaggg gaaaatgtag tacagagaaa 3660
gaacaggaag atctgctaaa gttccaaaga ttgaaaaacg cagtagaatt ccgggatgtt 3720
actgaatatg ctgaggttat taatgagctt ttaggacagt tgataagttg gtcatatctt 3780
agggagaggg atctattata tttccagctg ggattccatt acatgtgtct gaaaaacaaa 3840
tctttcaaac cggcagaata tgtggatatt cgtagaaata atggtacgat tatacataat 3900
gcgatacttt accagattgt ttcgatgtat attaatggac tggatttcta tagttgtgat 3960
aaagaaggga aaacgctcaa accaattgaa acaggaaagg gcgtaggaag taagatagga 4020
caatttataa agtattccca gtatttatac aatgatccgt catataagct tgagatctat 4080
aatgcaggat tagaagtttt tgaaaacatt gatgaacatg ataatattac agatcttaga 4140
aagtatgtgg atcattttaa gtattatgca tatggtaata aaatgagcct gcttgatctg 4200
tatagtgaat tcttcgatcg tttctttaca tatgatatga agtatcagaa gaatgtagtg 4260
aatgtgttgg agaatatcct tttaaggcat tttgtaattt tctatccgaa gtttggatca 4320
ggaaaaaaag atgttggaat tagggattgt aaaaaagaaa gagctcagat tgaaataagt 4380
gagcagagcc tcacatcgga agacttcatg tttaagcttg acgacaaagc aggagaagaa 4440
gcaaagaagt ttccggcaag ggatgaacgt tatctccaga caatagccaa gttgctctat 4500
tatcctaacg aaattgagga tatgaacaga ttcatgaaga aaggagaaac gataaataaa 4560
aaagttcagt ttaatagaaa aaagaagata accaggaaac aaaagaataa ttcatcaaac 4620
gaggtattgt cttcaactat gggttattta tttaagaaca ttaaattgta aaaaagattc 4680
gttgtagata attgataggt aaaagctgac cggagccttt ggctccggac agttgtatat 4740
aagaggatat taatgactga aaatgatttt tgttggaagt cagttttttc tgtggaaagc 4800
gaaatcgaat atgatgagta tgcatatggc agaagagctg tagaaggcga gaatacatat 4860
gattacatta ctaaggaaga aagaccggaa cttaatgacg aatatgtagc gagacgttgc 4920
attttcggta aaaaagcagg aaaaatatcc aggtcggatt ttagtaggat aagatctgcg 4980
ttggatcatg cgatgataaa taatacacat acagcatttg ccagatttat cactgaaaat 5040
ctgacgagac tcaatcacaa agaacatttt ctgaatgtga cacgtgcata ttctaaacct 5100
gattctgaaa aattgataca accgagatac tggcagtcgc ctgtagttcc aaaggataaa 5160
caaatatatt atagcaagaa tgcgattaaa aaatggtgtg gttacgaaga tgatattccg 5220
cctcgttctg tgatagttca gatgtgtcta ttgtggggga ctgatcatga agaggcagat 5280
catatccttc gcagttcagg atacgcggcg cttagtcctg ttgtacttcg agatcttatc 5340
tatatgtatt atctggatca tcaggatttg caaaaaaatg agttgatatg ggaagtaaaa 5400
aagcagttgg atcacttcga tttgacaaat agaaattatg atacaaatcc ttttgatgta 5460
gggggcagcg taaatgatca tatctgtgaa ctgagcgagc atatagcgaa ggctcattat 5520
atttatgaga gggctaagga aggaccattg caaaatgtaa ttcgggatat tttgggagat 5580
acacctgccc tttattctga aatggcattt cctcagctag catctataaa caggtgtgct 5640
tgcaattcgc tttcttcata tcaaaaaaat atttttgata ctgacatagc tatatatgca 5700
gatgaaaagg acacaagagg taaatcagac cgtatccttg ttgagggcgc atcttcgaaa 5760
tggtatgaat tgaagaaacg cgatgctaat aatgtcaaaa tttctgaaaa gctgagtata 5820
ctcaatacta ttcttaaatt taatagtgtt ttttgggaag aatgttacct tgatggaaat 5880
ataaaacaat cgagcggaaa gcgatctgag gcaggaaaaa ttctttatgg tcgcgacaac 5940
ggaaaagaaa atgtcggagt ttcaaaattg gaattggtgc ggtatatgat agctgcaggt 6000
caggaacaaa atctgggaaa ttacctggtg agttcaggat tttggagaaa aaatcatatg 6060
ctgtcattta tacaaggcaa tgatatagcg cttgatgaga tggatgaatt ggatctctta 6120
gactatattc tgatatatgc atggggattt agggaaaata tcattaaaaa gaacagtaat 6180
gtgaattctt tggatgaaaa gactagaaaa gtgcagtttc cgtttataaa gttactcatg 6240
gcaattgcaa gagatatcca gatacttata tgttcagcac atgaaaaaac agtcgatgag 6300
tcatctcgaa atgcagcaaa gaagatagat atattgggaa attatattcc ttttcagatt 6360
catcttcaga gaactaaaaa agatggtgga agagtggtaa tggatacatt gtgtgctgat 6420
tggattgcgg attatgaatg gtacattgat cttgagaaag gaacacttgg atgagcagtg 6480
atgaaaggat atttaaaaaa tttttggaaa aaggatcgat ttctgagcag aaaaagatgc 6540
ttttagaaga aaagaaatgt tcggataaac taactgcact gcttgggaat tactgcatac 6600
cgatagacaa tatttcagag tcagacggaa aaatatatgc ggtctataag cttccaaaaa 6660
atgttaaacc tttgtccgaa atcattaatg atgtatcctt ttctgattgt acgatgagag 6720
tacgtttgct tctcataaag agaattctgg aactcgtgtg tgcttttcac gaaaaaaaat 6780
ggtattgtct cagtatttca ccgggaatgc tcatggttga agattttgat ataccgatgg 6840
gaaatgtcgg aaaagtattg atatatgatt tcagaaatcc tgttccgttc gagtcagtaa 6900
atgaaagaca taattttaac gtttcaaata aatacacttc accggagctg ctcatccatt 6960
caagatatga cgagtcgaaa tctgtgagtg aaaaatcaga tttgtattct gttgcaaaaa 7020
ttgcggaaac aataatagga gattttaaca gtattattgc aaatggaaat ttgatactac 7080
ttgcaatgct tagagttttt atcagtacag ggaaaagtcc ggaacctgag tatcggtttg 7140
aatcgtcgga aaatatgctt tcagtatttg aaaatttgat caaagaaaat tgtttttttg 7200
aaaaaaacga ttatacatct atgtttcatc aggcgtatga caattttttt gaatggcagg 7260
aatgtttgat atcaccggat cacttggata aaaatatgtt cgaggcagct ttatcaaatc 7320
ttgaggatca gctgcttagg gttgatattg ataagtatag agcagagtac ttctataagc 7380
ttctccgaga gttgtctaat aaatataaaa atacaattac tgatgaacaa aaggtaaggt 7440
tggcaatact tggaatcaga gcgaaaaata atctgggaaa aagttttgat gcattggaaa 7500
tatatgagtc agtacgtgat ttagaaacta tgttggagga gatggcagag cttagtcctg 7560
tcattgcttc gacatatatg gattgctacc gatatgcaga tgcgcagaaa gtggcggaag 7620
aaaacattat caggcttcat aatagtaata ttcgtatgga gaaaaaaaga atactgcttg 7680
gaaggtcata tagttcaaaa gggtgcagca tggggtttca gcatattctt ggtgcggatg 7740
agtcatttga acaggcttta tatttcttta acgaaaagga caatttttgg aaagaaatat 7800
ttgagagcag aaatttagag gacagcgata gacttataaa gtctttacga agcaatacgc 7860
atattacgct gtttcattac atgcaatatg catgtgaaac aaggagaaag gaattatatg 7920
gagcactttc agacaaatat tttataggta aagaatggac agaaagactc aaagcatata 7980
taagcaacaa ggatatatgg aaaaactatt atgagatata tattctgcta aagggtattt 8040
attgcttcta tccagaagtc atgtgttcgt ctgcgtttta tgatgaaatc caaaaaatgt 8100
acgatcttga atttgaaaag gaaaaaatgt tttacccatt gagtctgata gaactgtatc 8160
ttgctctgat agagataaaa gttaatggga gtctgacgga gaatgccgag aagttgttta 8220
aacaggcatt gacacatgac aatgaagtca aaaaaggaaa tatgaatatt cagaccgcca 8280
tttggtatcg aatatatgca ctgtataacg atgtaaaaga tgaaactgat aagaataaaa 8340
ggcttttaaa acggcttatg attctttgcc gacgatttgg ttgggcggat atgtatagtg 8400
ctttggagaa ggatgggaag ttaattgatt ttttgagatt tgaggtatgt taaatgataa 8460
cacttgcatt agatgaaaat ggcaaatttg aagatgcttt ttctaaaaaa aatgaaaaac 8520
cgataatgat tgcggggata atctatgatg acaaggggaa agagtatgat gctgagaatg 8580
aacgctacag gatatccagt tatctgcgag cagtatgtga cagtttgggt gcgaaatacc 8640
ctcaggatct acattcaaat agtaatggaa ataaggcgac tgttgggaaa gtaaaatgta 8700
aaattggtga aacactaaag gaattcttga gagaaggaac ctatgaaaaa aaggaattgc 8760
cgacaaagaa cggttattta aataagagat ctggaaaata tgtaatgttt gcagaactca 8820
ggagtagtca gggagttaaa aagcgtgtta gtggttggaa tgacaatgat ctgactcagg 8880
atgaaaaggt cagcaatctg taccttcata tggcagaaaa tgccgttgtc agaatgctct 8940
tccataatcc tatatatgaa gatgtaacag atgtaaatct ctattttccc acgcgaaaag 9000
ttgttctgaa agatagagat agagaatacg ataaacaaga tttcaaaata tatggtgata 9060
aggacaagtg cgaagcagaa agcgggagat tggtgcatta tgatatcgtg tcatcggatt 9120
tttaccgtac gataatggag aacgaatgta caagaattaa taaaaagcaa ttaaatgttc 9180
attatatgaa cacaagccca atttcgtact gggagaaaaa tgaaaaatat aatacatttt 9240
tatatttggc tgacatagtt tgttctatgc tggattatta caaaaagggt tcgagtccgg 9300
cagagtggat ggattctttt gccgaatggg gaaacaaata ttttggtgat gatcagataa 9360
tcttatttgg gtatgatgat atagatgaca aatacatgga ggctgtagat gcagtaggac 9420
agggagagta ttttcatgcg ctggatatta tatatgatgc ggaatgtagt ggaagtgaat 9480
ttgagaagca ctacaaagat tattggtttc caaagcttat aaaaaagata cgaataacag 9540
caactgtgga taatttatgc agatcgatct cagatctgga gagttttaca tatcgaagta 9600
atcttgatca gcagaaactt ttgtggattt ttgaggaaat caaagctatc gtcgataagg 9660
gagattttgg aaagaaatat catacagatc aggttatgtt tgatatgtgt aatgccggta 9720
ttgctgtgta caatcatatc ggagattttg ggactgcaaa ggaatactat gatgagtgca 9780
tgaaacacac tggggatgtg gatctggtaa agatacttcg tgcatcaaat aaaatggtgg 9840
tctttcttga cgatgctttt aggtatggtg acgcgacaga acgtgccagg aagaatgttg 9900
aataccaaaa agctttgcac gatataaaga gtgagatttg tccggaaaag aaagatgaag 9960
acttgaacta tgccatatcg ctcagtcaat ttggacaggc gcttgcgtgt gaaaaaaatt 10020
ctgatgcaga gagtgttttc ctagagtcgt tgcggcatat gaggaaaggg actgccaatt 10080
atcagattac tctttcatat ttactccatt tttatctgga tatgggaatg acagattctt 10140
atcgagaaaa aacaaaggac tattttggaa gtgaaaaacc aaaggaacag ctgaaagaat 10200
tgctgaagtt atcgggaaag gatgatagta tagttacttt caaatttgca atgtatgtct 10260
atttacgtgc actttgggta ttacaggaac cgcttactga ttttatcaga acaagattag 10320
aggacatacg tgagactctt gtaaagaaga aaatgagtga acatatggtt ggacatccgt 10380
gggagttgat ttataaatat ctggcatttc ttttttatcg tgatggaaat tgtgaagctg 10440
ctgaaaaata tattcataaa agtgaagagt gcttggaaac acaaggactg actatagatg 10500
cgattattca taatggtaag tatgaatatg cagaattgtc aggtgacgag gagatgatgg 10560
caagagagaa agcgtacttt gatgaaaaag ggatagatag aaaaaatgtt tgtactttta 10620
tgtatcattg atgtttaata agatttgacc gaggagtgac aggtaatcgc cggtatatct 10680
ggtattacct gtcatttttt gatgaaataa gctacttttt gcctaaaaaa cgaaactgtt 10740
ggtgttttat gatgattgtg tcaacaaaag agagcaaaag aagaggagaa aagtaatgtc 10800
aatgatttca tgtccgaatt gtggtggaga gatatctgaa aggtcaaaga aatgtgttca 10860
ttgtggatat gtgttagtcg aagaagctaa agtagtgtgc acagaatgtg gaactgaggt 10920
agagagtggc gctgctgtat gtccgaagtg cggctgtcct gtaaatgata gtgagacgcc 10980
tcagaaagtt gaagtgacta gggtaaatgt atcttccgta atcagcaaaa aagtcgttgt 11040
aagcatactg atcgcagtga ttacaattgc aggttttttc tatggagtga agtattcgca 11100
ggaaaagaaa gcaattgaag agtcagtaaa gcagaaggaa gactatcaaa gtacgctaga 11160
gcttgcttcg ctaatgatgc ttcaaggagc ttcggatgca gaaacttgtg ggaatttggt 11220
taggaaagtg tggagcaact gcatttataa ggagagggat gaagaaaccg acaagtatac 11280
gtgtgatagc aggggtgcag gatggtttta tgatgatttt aatgatgcat taatggctct 11340
ttacagtgac agcagttttg gcaagaagat aaatgaaatc aaaaacggtc aggaaaccgt 11400
tgcggcgatg atgaaagatc tgaaaaatcc gccggatgag atggcagatg cctatgagga 11460
tattcaaaat ttttatgtgt cctatctaac gctgacagaa atggttgtga atccaactgg 11520
aagtttgagt tctttttcat ctgatttttc cgatgcggat acggaggtgt ccaatgccta 11580
tagccggatg aagttgtatt tagattaaac tattgaggaa aaaatggagg tgctttaatg 11640
cgggggagaa actgtggagg gtcatcaggc gacggactgc tggtacttct cgtactgctt 11700
gtcctttttt ataaaatcat gccattcata ggtttatgga ttttaatttt tggtgatgct 11760
gaacgtaaag atctgggtat gggtatgatt attgtcggga tagttctata tgtattatta 11820
gaggtttttt aatgtgagtt tctgtggtaa actataaaag tacaagcttt tgcgccgcac 11880
cgcataaata gcggatttat gaccattatt tggtgaaaaa aatggtgtac acctgtgttt 11940
ttttgttttg cgccgcaaaa tgcgccacgg aaccgcatgc agagcaccct gcaagagaca 12000
gggttatgaa aacagcccga catagagggc aatagacacg gggagaagtc atttaataag 12060
gccactgtta aaagttatga aaacagcccg acatagaggg caatagacat aaagaccaaa 12120
aacaggtcat ctgcatactg tgttatgaaa acagcccgat atagagggtg tgagagatat 12180
agttctcgtc acagtgcaga aaatgaccta ttatgtgccg aaaaacaaaa tgaaaaaaga 12240
atggaaaggc gtatttaatg aaatgctgat ctgttgattt gaattaacaa aaaaaggtcg 12300
ccccacggat gacaaaaaca tccgggggcg accctttt 12338
<210> 65
<211> 6098
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 65
tactgtgtgc ataagtcttc cttagatcca taggtacagc agttttattt attagcctta 60
gaaaatggaa aatagagctt ataaatgata tgatatttat gaataaaatg attgcattct 120
cgtgcaaact ttaaatatat tgattatatc ctttacattg gttgttttaa ttactattat 180
taagtaggaa tacgatatac ctctaaatga aagaggacta aaacccgcca aaagtatcag 240
aaaatgttat tgcagtaaga gactacctct atatgaaaga ggactaaaac ttttaacagt 300
ggccttatta aatgacttct gtaagagact acctctatat gaaagaggac taaaacgtct 360
aatgtggata agtataaaaa cgcttatcca tcatttaggt gttttatttt tttgtgatta 420
tatgtacaat agaagagaga aaaaaatcat tgaggtgaaa actatgagaa ttactaaagt 480
agaggttgat agaaaaaaag tactaatttc tagggataaa aacgggggca agttagttta 540
tgaaaatgaa atgcaagata atacagaaca aatcatgcat cacaaaaaaa gttcttttta 600
caaaagtgtg gtaaacaaaa ctatttgtcg tcctgaacaa aaacaaatga aaaaattagt 660
tcatggatta ttacaagaaa atagtcaaga aaaaataaaa gtttcagatg tcactaaact 720
taatatctca aatttcttaa atcatcgttt caaaaaaagt ttatattatt ttcctgaaaa 780
tagtcctgac aaaagcgaag aatacagaat agaaataaat ctctcccaat tgttagaaga 840
tagcttaaaa aaacagcaag ggacatttat atgttgggaa tcttttagca aagacatgga 900
attatacatt aattgggcgg aaaattatat ttcatcaaaa acgaagctaa taaaaaaatc 960
cattcgaaac aatagaattc aatctactga atcaagaagt ggacaactaa tggatagata 1020
tatgaaagac attttaaata aaaacaaacc tttcgatatc caatcagtta gcgaaaagta 1080
ccaacttgaa aaattgacta gtgctttaaa agctactttt aaagaagcga agaaaaacga 1140
caaagagatt aactataagc ttaagtccac tctccaaaac catgaaagac aaataataga 1200
agaattgaag gaaaattccg aactgaacca atttaatata gaaataagaa aacatcttga 1260
aacttatttt cctattaaga aaacaaacag aaaagttgga gatataagga atttagaaat 1320
aggagaaatc caaaaaatag taaatcatcg gttgaaaaat aaaatagttc aacgcattct 1380
ccaagaaggg aaattagctt cttatgagat tgaatcaaca gttaactcta attccttaca 1440
aaaaattaaa attgaagaag catttgcctt aaagtttatc aatgcttgtt tatttgcttc 1500
taacaattta aggaatatgg tatatcctgt ttgcaaaaag gatatattaa tgataggtga 1560
atttaaaaat agttttaaag aaataaaaca caaaaaattc attcgtcaat ggtcgcaatt 1620
cttctctcaa gaaataactg ttgatgacat tgaattagct tcatgggggc tgagaggagc 1680
cattgcacca ataagaaatg aaataattca tttaaagaag catagctgga aaaaattttt 1740
taataaccct actttcaaag tgaaaaaaag taaaataata aatgggaaaa cgaaagatgt 1800
tacatctgaa ttcctttata aagaaacttt atttaaggat tatttctata gtgagttaga 1860
ttctgttcca gaattgatta ttaataaaat ggaaagtagc aaaattttag attattattc 1920
cagtgaccag cttaaccaag tttttacaat tccgaatttc gaattatctt tactgacttc 1980
ggccgttccc tttgcaccta gctttaaacg agtttatttg aaaggctttg attatcagaa 2040
tcaagatgaa gcacaaccgg attataatct taaattaaat atctataacg aaaaagcctt 2100
taattcggag gcatttcagg cgcaatattc attatttaaa atggtttatt atcaagtctt 2160
tttaccgcaa ttcactacaa ataacgattt atttaagtca agtgtggatt ttattttaac 2220
attaaacaaa gaacggaaag gttacgccaa agcatttcaa gatattcgaa agatgaataa 2280
agatgaaaag ccctcagaat atatgagtta cattcagagt caattaatgc tctatcaaaa 2340
aaagcaagaa gaaaaagaga aaattaatca ttttgaaaaa tttataaatc aagtgtttat 2400
taaaggtttc aattctttta tagaaaagaa tagattaacc tatatttgcc atccaaccaa 2460
aaacacagtg ccagaaaatg ataatataga aatacctttc cacacggata tggatgattc 2520
caatattgca ttttggctta tgtgtaaatt attagatgct aaacaactta gcgaattacg 2580
taatgaaatg ataaaattca gttgttcctt acaatcaact gaagaaataa gcacatttac 2640
caaggcgcga gaagtgattg gtttagctct tttaaatggc gaaaaaggat gtaatgattg 2700
gaaagaactt tttgatgata aagaagcttg gaaaaagaac atgtccttat atgtttccga 2760
ggaattgctt caatcattgc cgtacacaca agaagatggt caaacacctg taattaatcg 2820
aagtatcgat ttagtaaaaa aatacggtac agaaacaata ctagagaaat tattttcctc 2880
ctcagatgat tataaagttt cagctaaaga tatcgcaaaa ttacatgaat atgatgtaac 2940
ggagaaaata gcacagcaag agagtctaca taagcaatgg atagaaaagc ccggtttagc 3000
ccgtgactca gcatggacaa aaaaatacca aaatgtgatt aatgatatta gtaattacca 3060
atgggctaag acaaaggtcg aattaacaca agtaaggcat cttcatcaat taactattga 3120
tttgctttca aggttagcag gatatatgtc tatcgctgac cgtgatttcc agttttctag 3180
taattatatt ttagaaagag agaactctga gtatagagtt acaagttgga tattattaag 3240
tgaaaataaa aataaaaata aatataacga ctacgaattg tataatctaa aaaatgcctc 3300
tataaaagta tcatcaaaaa atgatcccca gttaaaagtt gatcttaagc aattacgatt 3360
aaccttagag tacttagaac tttttgataa ccgattgaaa gaaaaacgaa ataacatttc 3420
acattttaat taccttaacg gacagttagg gaactctatt ttagaattat ttgacgatgc 3480
tcgagatgta ctttcctatg atcgtaaact aaagaatgcg gtgtctaaat ctttgaaaga 3540
aattttaagc tctcatggaa tggaagtgac atttaaacca ctatatcaaa ccaatcatca 3600
tttaaaaatt gataaactcc aacctaaaaa aatacaccac ttaggtgaaa aaagtactgt 3660
ttcttcaaat caagtttcta atgaatactg tcaactagta agaacgctat taacgatgaa 3720
gtaattcttt taaagcacat taattacctc taaatgaaaa gaggactaaa actgaaagag 3780
gactaaaaca ccagatgtgg ataactatat tagtggctat taaaaattcg tcgatattag 3840
agaggaaact ttagatgaag atgaaatgga aattaaaaga aaatgacgtt cgcaaagggg 3900
tggtggtcat tgagtaaaat tgacatcgga gaagtaaccc actttttaca aggtctaaag 3960
aaaagtaacg aaaacgcccg aaaaatgata gaagacattc aatcggctgt caaagcctac 4020
gctgatgata caactttaaa aggaaaagca gtggattctt cacaaagata ctttgatgaa 4080
acgtatactg ttatttgtaa aagtatcata gaagcattag atgaaagcga agagagatta 4140
caacaatata ttcatgattt tggagatcaa gtggattctt cacctaacgc acgaattgat 4200
gcggaattac tacaagaagc aatgagtagg ttagctgaca taaagcggaa gcaagaagca 4260
cttatgcaat ccttatcttc ttctacagca acgctttacg aaggcaagca acaagcgtta 4320
cacactcaat tcacggatgc gctggagcaa gaaaaaatat tggaacgcta tattactttt 4380
gaacaaactc acgggaattt ttttgactca tttggagaac ttgtctatcg aacgggacaa 4440
gcagtgcgtg aattagctaa taacgtcaca ttcgagagcc aaacaggaag ctatcatttt 4500
gataaaatag atgcttctag attccaaact ttgcaagaaa tgttgccaaa ggcaaagaaa 4560
aaagcattta attttaatga ctaccaaata acatggaatg gcaccacgca ccttttatgg 4620
aaaaatggta aagtggatgc agaagcaacc aaagcttata acgaggcgaa actgaatgga 4680
aagctaccaa aggaaggtaa tgtagcaaca caagatgcag aactattaaa aggcattttg 4740
gcttcactga aaaacaagaa agatcctatc actggagcag atataagcag tgtgcatgta 4800
ttatctatcc ttagcgggct cgcattctcc tatacagctg ggaattataa gggaagaaaa 4860
cttactgttc caaaaagttt cttagacaaa ttaaagaaaa accgaaaatc taaagtacct 4920
aaactatcta gtttatcaga aaaacaacaa ctaaaactcg caaataaata caagaaaaaa 4980
tcacctattc caattccaga tgatgctaaa atcaaagctc agacgaaaaa ggctggttat 5040
gaacaaatat cttataaatg gaaagagaat gggataacct ttgaagttag atggcatact 5100
aggacaccag gtgcaccaaa ggaacaagga aatacgtttg ttatagaaag aaaaattcag 5160
ggtacagcag aagggaaaac aaaagttcaa caaatattgg ttggagataa taagtgggtg 5220
agtaaaagtg agtggcaaaa ggctataact gataagaaaa atggtgtaag tacctcggag 5280
caaaataaaa tgttgtctga tggacattgg aaagaataga aaggagcaaa atgatggaag 5340
attattataa aggttttgag ggatatccag agatagattt ttatacgtat atagatgata 5400
tgaaattggg tatagcaatg tgggaaggat actttgacaa cattatgaaa gaaattaatc 5460
caagtaacgg aagatggact tcattagcgt attattatca tttagatgag gggtggtatg 5520
atgaaagtcc ttgggaaata ccaagtaata cagaagcatt agaattattg gaaacaatcc 5580
atatatctaa tctagatact atcacacaag agatattact taaattaata aatttattaa 5640
agaagaatat aaatagacaa gtttatattg aatactcata aaaaagatga ttatgatata 5700
ttatagaaca aacgaacaag ccccaaatac gaggtttgtt cgtttgtttt caatataatt 5760
atttgccacc aagtgagata ttacggtttt aaatagctta tttgacgata ccaaaccctg 5820
ataagagaaa gaagaaagag aaagctggtg tagttgtttt aagtgaacta gataaaaaat 5880
taatagcaaa acttgaaaaa gatggtgtga aaatatcaaa agaagatgtt ataggaataa 5940
aataattgcc agatgatgag aaatcgtttg gctggaaaaa ggaaatccat ccgctggatt 6000
tgagcatatt cttattgaac atggtgaaca atttgctaaa tagggaattt caaaagctga 6060
gttacctgat tttttgatga ctgctttaga aaaggaaa 6098
<210> 66
<211> 6222
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 66
attctttaaa aatatctaat aatttattta ctatatactc taatacatct tttaacctat 60
ctaaaacatc atcacctaca acatcccaaa aatcatctaa aaagttaaaa aaatccatct 120
ttatcaactc ctatatctat tttttattgt gtaattcctg agttacaaaa ccattataac 180
acgtattaca cacgtagtca atacttcaaa aaaatttttt gtatattttt ttgaataagt 240
aaataaaaag agctgtgtag ctctttatta aaatcaatat ttttattttg ttaacaaact 300
tagacaacat taaatttaga aacctatata tatttcagta cttttcattt ttaggtagtc 360
taaatcagaa atggttttgt ctaaatgatg tatgtaagtt ttagtcccct tcgtttttag 420
ggtagtctaa atcagaagtc atttaataag gccactgtta aaagttttag tccccttcgt 480
ttttagggta gtctaaatcc catccaaatt atgggataat atgttacttt ttattttaat 540
atttgattat ttattgtttt tttactgatt tagattaccc ctttaattta ttttaccata 600
tttttctcat aatgcaaact aatattccaa aatttttgtt tcttttctta tgatcttttc 660
tccgatagtt atttctccag ataagatttt catttttttg aattgatctt ctgttagaat 720
taatgttctt actgatgaat tttctggaac tatcattgac aactgatttt cataggaaat 780
tattttttct tttgtgctag aacttacaat gtatactgat ttttgtacct gataatatcc 840
ttttcttata atttcttttc taaattttgc atattctttt ttttcttttc ctgtttgcat 900
tggaaaatca tacattagaa tccctacata attagtactc ataatcctct atccttaact 960
caggaatttc tacttctgac atttctcctg taaaataatt tctaatatta tctaaaaaat 1020
aatcaatcac ttgagccaat tcatattttt tatttttcca ataaactttt tgtgttaata 1080
ccaataacaa tttttgtctt aatgatttat tcaaacttac ttcttcctgt tgattaaaat 1140
atacgatata atctaccatt ggacgaaata tttcaataat atcatctgca aaattataat 1200
tattaaattg tgaactgtga tgtattccca aacttggatg aaatccttta gccacaattt 1260
ttgaagagat taagcttctc aaaaccatat acccataatt taatgccgaa tttgtcccgt 1320
cttcaccaaa tctcttaaat tttttcccaa aaagttcacc aaaatacatt cttgcagcaa 1380
ttgcttcctg atgttccgct tcttttcctt ttaatctaat attattttca tatgcttcca 1440
acttatatga tacttcctga gattttttca aaaactgcaa taaatttctt tgattttcta 1500
tttttctcat tacaattttt ctccagattt cttctttttt atcgtcaatc cagctcactt 1560
gctcattaat tcttgttgtt acttgaaaat gattatacag tcctaatgaa tgtaaaactg 1620
gctgatgttt ttcattacaa attatcagtg gaatattatg ttctgataat cttaactgta 1680
atattccgct aattttacat ctgcaatttt caactacaat tgccatgata tcatttaaag 1740
atactttatc agccttattt tcatcatctt catttatcat cacaagctgg ttatttaaaa 1800
ctgataattc attgactctt gttacatgga taatattaga catttttatt actcctttac 1860
tctaaagctt tatattcaaa cataactttc acaagttcac acaattcttc tgaatttcta 1920
tcagtcatta attttttctt ttttaaattt ttcaaatgta caattttttc cgattctaaa 1980
gtctgaattt ctattttctt atctgctcct attttaaatg ttgctacaaa accatattcc 2040
tttaatatat ccactattga tttcataatt gcatttttaa gttttctatc ataagaaagt 2100
aattttctta aattttccag cacttctaaa agtgaaattt cagcatgcgg aatatagtta 2160
aaatgtgcaa tatagtttcg tatatacaaa tcttttttct cttgttttaa tttttttact 2220
tttttatcag aatagatgct tcttttttct acattatctt tgtataattc tttataaaaa 2280
tttatatatt tttcaacaat ttgcccactt ttatatttta catttttact gttatcaaaa 2340
ttaaatattt cttcaatata atgattttca ggaaattcac ctttcaatct aaatcttaag 2400
tccctttccc agatcgaagt atatcccaca agtctgtgga gtatttttaa taacaagcct 2460
tgcaacaagt ttaattcatt aaattccact ttatttttca aatgagtata tttttgtata 2520
tttccaattg ctttttcata ttctttataa tcttcatcat taaatttttc atctttttta 2580
ggtcttgcat attttctatg taaattttgc tgcattgtat aatttttttc tatttcattt 2640
tttttattgc tgtattcttt caattctttt aaacttattt tatacttcgc tttatcagct 2700
attttttcaa gtaaatttaa catcccatat ttttttatat tataaaaagc tctatgcttt 2760
ataatatttt ctccatcaaa atatatttta tttgtgtcaa atttcttcaa ttctttccta 2820
tcttttattt tattttcatt aaaatctaaa aattttccaa tttcattcgc ttctaattca 2880
aaatcttctg ttactctatt attatctaaa tttaaaagat ttataagttc aagttcatct 2940
gaaaaagttt cttctttatt tgcactctga tatttttcaa gacttccctt caaattagtc 3000
aattctttat gattaagcaa ttttaaaatt aaataaaaca tattcaaatt ttcagtgtat 3060
tttaatatct ttcctaattt tatctctctt acaaattcat ttatttcatg tggaatttct 3120
ttattcctat tatgtttttc ataatttttt aaaattttat catatttttc tttattatct 3180
ttttttattt ttattttaga aaatatatca ttattatcat tgttattatt actttctata 3240
tattttaaat tatttttatt caaataatct ataaaacctt ttaaaaatat ttgttgtata 3300
aaatcaatgt atgtattttt ttcttcttta tcttgattat taatcatctc cctactttgt 3360
ataatagcaa gatattctac tggtacagtt ttttctatat tttcaaattt ttgatattta 3420
taatgtcctg ttttttgatt tctttgttta tttattttta ttacttcatt agttatttta 3480
aaaaaaactt tactattttt aacaaattta ttaagaaatt caccataata aatatttttc 3540
aaaagatata tttgagcatc tttttcttct ttatccttag gaacactcca aaaaaatttt 3600
aaagtatttc ttaaatcttc tattttatta tataatttcg taaaagaagg aacaaaagga 3660
atattcttat ttacaaaatt aaattttgta ttttttaaat atttaattat cacatccttt 3720
tcataataat taaatacatt tgcactattt aactgcttaa atatcttcaa tttcaatttt 3780
ttctcattta tttcattttg aaacattttt tttgaaattt cagaaggagc tatattttta 3840
aatgcaaata tatctttccc ttctaattcc aaattaaaat gcacaatccc atgtctaata 3900
ctgctaatag cttcatcaat atttgcaaaa aaatcttcta tctcattttt attatccata 3960
ttaaaatcat aactatagaa catttttaaa ttttctttta cttcattttg cttgttttca 4020
ttatatattt tatcaacttc tccagaaaca tatttttctt cgcccttatt attttttaca 4080
gtttttcctc tcattctacc tgtaatatca ttctcatttt cagtttcaag aatatttctc 4140
aatgaaaaat atgcaaccga agaaactcca attatatttc gtaaaaatgc ttcattttgt 4200
ctattcctag caataaaatc acttgttgca atctctccaa cttgtaaata ataattgtat 4260
ttcccacaat ttcttacata agtatccaat ttatttagta atttgttttc aattaatttt 4320
tttaaatttt gatattcaaa tattctctta attttatcgt tacttatgtt actcagtctt 4380
ttatacacat aatttttcaa aagctgactc atttcaattt ccacaaaatg acaaaaagca 4440
tattttatat ttttatcatt aagttcttct ttatccaaat aatatttata aaacacttgt 4500
gattttttta attcactcat atccggaatt ttttcaatta attcttttat attatttaca 4560
ttttgtattt cttcgtaaat aattttagca aaattttctt tatcattttt tcttccaatt 4620
attttgtgat agtattctct tattttatat ttttcatgtt tttttgaatt ttctattaaa 4680
aaaaataact tctcaatatc ttctttttta tacaatttat caaatgcttc ctgtacatta 4740
tttatataat cattacgctt tgctgattct ctataataat cataaataat atttcttttg 4800
ctcttccctc caactttttc aacattattt tcattaattt tctgataatt agccttattt 4860
tcttcaaatg aatattttaa agaatttatc ttattcaatt ttgcctcaac atcttttcta 4920
aatatttcta attcttcaga gttcacatct tcatttaaca atattttctt taaaactgaa 4980
aaactatttt tattttttaa atcatattct gaaatatctt cttcagaata atttttatcc 5040
tgtactgcat ttttctcttt cctattcttt aaatacagaa cactatcttt tagatgcaat 5100
actttatttg aaaaaaactt ttttaaattt tctcttctta ttctattttc ttcttcactt 5160
gcattatcag gattttttat atatatatcc agtcttatac ttaaaagctc tgacaatctc 5220
tcactagtcc tattttcttc gctcgtactt tttactaatt ttccctcttc aatatatttt 5280
ttatgcgaaa ttccatcaac ttttgtaact ttcatatata aaaacctcct aatatctata 5340
ttttttactc aatacctaat tcttttttca atgctttttg taaaatttgt gaaaaattca 5400
gatttttttc ctgtgccaat atatctaacc aaacaggaat tgttaaagtt ttctttttaa 5460
gtgcatttgt aacttttgcc acttcataca ctggatcaac agataaaata tacaaatact 5520
gattttcttt cagtttcaca tcctccactt ttgaaggctc aggaaatttt tttcttacat 5580
ccaaaaaatc agccaaatgc agacccaatg tctctctcaa attggaaaca gcctcctcca 5640
tgctatctcc aaatgtagca taataattta tctctccatc ttcaaactta tcaaaatcaa 5700
caatacaacc ataataagtc ccatcttcct tagttaccac tgctggataa aatacatcca 5760
ttttaattat ctccaatcta taccacgtgt taaatacgtg tttaaaaata tttataaaat 5820
tttttagcat ctctgctaaa ataaaacaat tatttcaaat ttttctattc cttaatcact 5880
cattgttagt gattcttttt ttacttggac aatttttcat ttaatttctt caattttttt 5940
aaaatcacat ttttttaata ttccttattt aattgcaaat tttcattact tttggggtgc 6000
tctaaatccc atccaaatta tgggataata atttttagtg aaagcaagaa gggactagaa 6060
tttaatccca acttgttttt caatacttct taatgttcct acaggtatat cttttgaata 6120
tggtactgtg accacacctt ccacacctgg gatcatccat tgataatgac tacctcttat 6180
acgcacaact tttccgccta attttctaaa tcttttttcg at 6222
<210> 67
<211> 6337
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 67
ctttctatct ttttcaaata aaattaggct ctagttagcc taatcgcata attatttatt 60
atagtataat tcttattttt tttcaaccta aaaatttaaa acatctccaa aaattttcgt 120
ttcagaacaa ccaagcaacc atattcaaaa aacaataaaa aatgagcaag aattgaaatt 180
ttattctcac tcagaagtta tttttattaa atatcacttt tcgatattgg ggtggtctat 240
atcaatttaa aagacagaat agataattct ttagagtttt agtccccttc gatattgggg 300
tggtctatat cagaagtcat ttaataaggc cactgttaaa agttttagtc cccttcgata 360
ttggggtggt ctatatccca tcctaatttc ttgctgatga gatatttatt tctaattttt 420
ctattttgtc tttattttca atactttcaa tcctattttt ctctttatta ataatataga 480
accaccctat actattatac catatttttt gatttttcaa aattccaata ttttgttttg 540
tgaaattttt tctcccattg tcacttctcc tgcaagtacc ttcatttttt gaaactgatc 600
ttctgtcagg ataatggaac ggattgatga attttctgga gcgagcattg ataactgttt 660
ttctgccagt tcgatttttt cttttgtttt cgacctcatt atatataccg atttttgaag 720
ctgataatat cccttttcta tcaatttttt cctaaaagtc ctatattcaa atctctcaac 780
atctgtctgc ataggaaaat catacataag cagaccaaaa tactcaatac tcatagtcca 840
tcacgctcaa tgtcggaatt atcacttctt catcttttac aaaataattt cgtatactat 900
ccaaataata gtctaccgct tggaaaaaat catatttctt attgttaaat aataccttct 960
gctgtgctac aagaagtatt ttttgcctta tttccttact taatttcact tcattcaaaa 1020
tatccttgta catataaaca agataatcca ccataggacg aaaaacctct attatatcat 1080
cagaaaaatt ataggcatta aactgtgact tatgatgtaa tcctaaactt ggatgaaatc 1140
cttttgctac aatctttgat gatattatag ctcttaaaat catatatcca taattaagtg 1200
cagaattcac tccatcttca tcaaatcttt taaaactatt actatacaat tcctgaaaat 1260
atatccttga agctattgct tcctgatgtt ctgcactcgc atcatctttt ttcaagtttt 1320
ccttatatgt tttcagtctt tcaatggaaa tatcactttt ttcaagatac tctaacaatg 1380
ctctttgatt ttcaatctta ttctccacta tcctgctcca caatttttcc tttttctctt 1440
tttcccactc aatctgctca tttattcgta aagtcacttg aaaatgatta aataatccca 1500
gcgaatgaat ttcaggctga tgtttctcgt tgcaaataat aatcggaatg ttattttcca 1560
ccagcctcaa ctgcaaaatc gcactaatct tacaatagca gttttcaata actatcgcag 1620
atatatcatt caaagaaatc ttatttttct catcattatt gtcttcatca accattataa 1680
gctgattatt cgatattgac aaatcatcag cccttgttat gtgaattata ttgggcattt 1740
taatcatact ccttataaat ttcattctta taacgtatca ttcgtatttt ctatttttgt 1800
taaaagttct attatcaagt ttttaatata atcagaatta taactttcta attctaaaac 1860
agaaactttt ttaggtttca ttaatctttc aagtatatca ttattaccga taagtttaaa 1920
ttttttcttt aattcatcat aatctaaatt cacatctttt ttaaatactt caaatacact 1980
tgcataagtt gaattattat aacgtgtact atatgataat aaattagaaa ctctatcaat 2040
ttgttctgca atactgtaat cagcaaacgg atttcttaca atatagaaat gtgaaatata 2100
gtttctaata ctttcatttt ccggcttatt aatttcagaa ttttcagaca aatcaattcc 2160
aaatccataa catattttct caaatttttt ataagattct tcatcaaaaa atttatagta 2220
tgctgttgtt gtataaaagc catcagatcc attacgctta ggataagctc tacttattcc 2280
agtattgtag ccacttaact taataattcc taattctctt agcccattta caatatagtg 2340
catatctctt tcaaatctag ccatttgaat agcaagtttc caatttatat ctatcaaata 2400
actttctatt ttattcaaat aattaaattc taccaaatct ctaatttttt tgtattcaga 2460
aactctatta taatcttttt caaatgattt atagttttta ttttgtatat tttttgcaaa 2520
aaagtcatca ttttctttca atttttttat atacttctct ttgtattctt tagaatatcc 2580
atttagttta tcatttagat ttttcaatat tgcatcaatt tcagatattt tattttttct 2640
aatattttta ccatcaatat taaataaaaa ttttgcatca gccattttaa tatcatttga 2700
aattaatcca taaattttat caaaatttgg atttccaata tttaaaaata aattcttttt 2760
ataaatatat aattcattct tacgttcttt aggataatat atttcttgaa atttattttc 2820
attctctgat tccatatctt ctattaaatt atctatttct tttttgtatt tttttaaaaa 2880
atcagaatta aatattattc tacacaatat tttactcttt atttcctgat ctttatcttt 2940
tatatactga tcaacctttt ttttcaaatc ctttttattt atgtttgata actttctttg 3000
ttcatcttgt aatatattcg attttttatc tatctcaaat ttagtttcat catcaaaaat 3060
tacaattttt tctaattttt tctctaaaac atcacaacca ttaatatcat ctttaaattc 3120
agttaatata ttatttttta tatcctcata ataattatta aaaatttctt ttttagtttg 3180
tattttaaaa tcatcaaagt ctttttctat ctctttcatt ttttgaataa attcttctaa 3240
attaagattc caattttcag ttatacattc atttctcaaa gtatttaatt gcattatttc 3300
atctaaaata tctataatat tttgatattc tgaagtattt aaccaaactg atgttgcaaa 3360
aaatctattt ctaattttat ttataaccgc attactattt aacagtgcaa atattgaaat 3420
tatatattca aaatcatcat ttattactat agttttatca ctagtcttta cagttattct 3480
ttcgtaagtt ttattatcat taatgtcttt tatttgtttc ttaatttctt gaatattcat 3540
tttaaaatct gaaaaatcaa aaagttcctc ataatttttt ctcaaatatc caatataaca 3600
ttctattact tttttctgat attttttaat agctttatta ttaccttttg aagcagaaat 3660
ctgagcattt ttataataat tttctataat attttcatct atttcatcaa tgtttcctaa 3720
agttttcttt aattcttgta aaaatatatt cttactttca ttttcttcta aatcatcttc 3780
taaaattaat ttcttataca attctttatt cacatatatt aaagcattta atactatttt 3840
ttctgtttct atagtatcaa atggttcatt cttaggatta ttcctatata aatttaatat 3900
ttcaggaagt actttagaaa aggatggtaa atatttaata tcattattat tttcttctga 3960
aattttaata tcatttattt tagtaattat atttttttta tctttaaata ctacatctaa 4020
atttaatgct tttgacactt cttcatctga tatttttaaa ttttgaatta tatttatgac 4080
tttattatag tcatcttgcg ttccttgtaa atctctttcc ttgctaatcg catgtaatat 4140
cctgtttctt tcatttgttc ctatctttgt aaatttccta ataaaattat ttgtaatgtt 4200
atttttatta tctataaaat ctaagtctct tattattttt atttttgaat ttaaaatttt 4260
tttatcaagt acgtaatttt tttctcgatc tcctccaaag aaatctatat tttcatcatt 4320
atttatattt tctctagaaa aaatcttatt taattccata ttggtagaag caaaaaaagt 4380
aatcaattct aaatccaatt cctctttagc gtgaagtcta gaaaaatcat cagtatttac 4440
tgttgtcata tctatatcat tatgtcttaa tttccctaaa tacataatat gctctaacgt 4500
atattgctta actcttttta aaattttttc agataatata ctttcattta aaattttttc 4560
tatttctatt ttttccattt tctttaatct gactttttgt tcatttacca atattttttc 4620
aattcttcct ttcaaatatc gatatatgat tttatatagt tctttttctt catcagattt 4680
ctttgaaaat tttttcgaat caaaattaac tttataatgt tttttaaata ttccaaaaat 4740
ttctgtatca caatttcctt tttttagttc tttttctaat ttttttatta attcatctat 4800
tttaaattct gctaaaattt tttctatttt ttcttttata ctattatttt ttatattttc 4860
tacaaaaaat tttacaattt tatctttttt attttctctt tctattttaa atttttcgtg 4920
cttatctaat agtacataag attttatata tgttctattt cttctctttt caagaaattc 4980
attattaact ttttttactt tttcaattct tttagtaata ttccaaaatt ctaactcttt 5040
tataacaaaa tcagctatat cttctactgt taaatctaca tttatattta aaattttttc 5100
aacaagcatt tttttatttt tagatttctt tttatcacca ccaacattaa gataaaattt 5160
tacaaaaccc agaatttcta aattactttt tattttttct cttatttcca taaaattagt 5220
caaaataaca tctattttat catctttcaa taatttttct cttaaatgtt cttcataata 5280
tcgattttca aatacttttt ctgtttcatt ttcaattatt ttttctataa tcttatataa 5340
actcatgtta atatttttaa aaatttcgta aattgatttt tttgtttcta attcatcatt 5400
ttctattatt cttaatatta ttgaacaatc atttagtgtt ttattagtat actcatctct 5460
gatatctatc tctatttctt cttcattctc ttgtctcttt atttctattt ttttatcatc 5520
tttagttatt ccttgcctaa ttgcttcatc tattattttc ttttttgtaa tccccaatgc 5580
tttcaatttc tcagattttc catatgcttc tatatataat acaacttctt ctgtttccaa 5640
aaaatcatca ttattttcta ttcttatgat tccttcttta cctttcaact taaatagaat 5700
atttcctgca tgaaattttc ttgtaaattc tttaagaata ttatcatttt ttttgtaatt 5760
aatatatttt ctaataaatt tattattatc aattttttct ttattattat tttcattaat 5820
atttaaaatg tatttgtttc catcatagtt ccttttaact tttactttcc gttttatttt 5880
aaaatctttt ttatcacgaa cttcatacca tctcttatgt ccaaataaat ttcccattcc 5940
aatctcctcg tttctacttt aatctaataa aatattttta aattaaatca attttacatc 6000
tttctaatca aaaatacaat tttccatttt tagtatacca catcaatatt aaatctcaaa 6060
aaaataagga gccgtcaaac atagctccct acttctattt actcataatc cccatctatc 6120
cttacttttc gtaaaatcaa tccttctttc gcctttagat ccaacttaat tttcccattt 6180
gaacctgttc taaatgttct gccttctgtt accaaatcaa taaatctttc atcctgataa 6240
tttgtttcaa attccacatt ttcccagctg ttaaacgaat tatttattac aacaataatt 6300
aaatgatcct cgattactct ttcatacaca attattt 6337
<210> 68
<211> 173
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 68
aatatggatt acttggtaga acagcaatct acgccagtga attcgagctc ggtacccggg 60
gatcctctag agtcgacctg caggcatgca agcttggcgt aatcatggtc atagctgttt 120
cctgtgttta tccgctcaca attccacaca acatacgagc cggaagcata aag 173
<210> 69
<211> 173
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 69
ggccagtgaa ttcgagctcg gtacccgggg atcctctaga aatatggatt acttggtaga 60
acagcaatct actcgacctg caggcatgca agcttggcgt aatcatggtc atagctgttt 120
cctgtgttta tccgctcaca attccacaca acatacgagc cggaagcata aag 173
<210> 70
<211> 56
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 70
ccaccccaat atcgaagggg actaaaacta gattgctgtt ctaccaagta atccat 56
<210> 71
<211> 1434
<212> PRT
<213> Leptotrichia shahii
<400> 71
Met Gly Asn Leu Phe Gly His Lys Arg Trp Tyr Glu Val Arg Asp Lys
1 5 10 15
Lys Asp Phe Lys Ile Lys Arg Lys Val Lys Val Lys Arg Asn Tyr Asp
20 25 30
Gly Asn Lys Tyr Ile Leu Asn Ile Asn Glu Asn Asn Asn Lys Glu Lys
35 40 45
Ile Asp Asn Asn Lys Phe Ile Arg Lys Tyr Ile Asn Tyr Lys Lys Asn
50 55 60
Asp Asn Ile Leu Lys Glu Phe Thr Arg Lys Phe His Ala Gly Asn Ile
65 70 75 80
Leu Phe Lys Leu Lys Gly Lys Glu Gly Ile Ile Arg Ile Glu Asn Asn
85 90 95
Asp Asp Phe Leu Glu Thr Glu Glu Val Val Leu Tyr Ile Glu Ala Tyr
100 105 110
Gly Lys Ser Glu Lys Leu Lys Ala Leu Gly Ile Thr Lys Lys Lys Ile
115 120 125
Ile Asp Glu Ala Ile Arg Gln Gly Ile Thr Lys Asp Asp Lys Lys Ile
130 135 140
Glu Ile Lys Arg Gln Glu Asn Glu Glu Glu Ile Glu Ile Asp Ile Arg
145 150 155 160
Asp Glu Tyr Thr Asn Lys Thr Leu Asn Asp Cys Ser Ile Ile Leu Arg
165 170 175
Ile Ile Glu Asn Asp Glu Leu Glu Thr Lys Lys Ser Ile Tyr Glu Ile
180 185 190
Phe Lys Asn Ile Asn Met Ser Leu Tyr Lys Ile Ile Glu Lys Ile Ile
195 200 205
Glu Asn Glu Thr Glu Lys Val Phe Glu Asn Arg Tyr Tyr Glu Glu His
210 215 220
Leu Arg Glu Lys Leu Leu Lys Asp Asp Lys Ile Asp Val Ile Leu Thr
225 230 235 240
Asn Phe Met Glu Ile Arg Glu Lys Ile Lys Ser Asn Leu Glu Ile Leu
245 250 255
Gly Phe Val Lys Phe Tyr Leu Asn Val Gly Gly Asp Lys Lys Lys Ser
260 265 270
Lys Asn Lys Lys Met Leu Val Glu Lys Ile Leu Asn Ile Asn Val Asp
275 280 285
Leu Thr Val Glu Asp Ile Ala Asp Phe Val Ile Lys Glu Leu Glu Phe
290 295 300
Trp Asn Ile Thr Lys Arg Ile Glu Lys Val Lys Lys Val Asn Asn Glu
305 310 315 320
Phe Leu Glu Lys Arg Arg Asn Arg Thr Tyr Ile Lys Ser Tyr Val Leu
325 330 335
Leu Asp Lys His Glu Lys Phe Lys Ile Glu Arg Glu Asn Lys Lys Asp
340 345 350
Lys Ile Val Lys Phe Phe Val Glu Asn Ile Lys Asn Asn Ser Ile Lys
355 360 365
Glu Lys Ile Glu Lys Ile Leu Ala Glu Phe Lys Ile Asp Glu Leu Ile
370 375 380
Lys Lys Leu Glu Lys Glu Leu Lys Lys Gly Asn Cys Asp Thr Glu Ile
385 390 395 400
Phe Gly Ile Phe Lys Lys His Tyr Lys Val Asn Phe Asp Ser Lys Lys
405 410 415
Phe Ser Lys Lys Ser Asp Glu Glu Lys Glu Leu Tyr Lys Ile Ile Tyr
420 425 430
Arg Tyr Leu Lys Gly Arg Ile Glu Lys Ile Leu Val Asn Glu Gln Lys
435 440 445
Val Arg Leu Lys Lys Met Glu Lys Ile Glu Ile Glu Lys Ile Leu Asn
450 455 460
Glu Ser Ile Leu Ser Glu Lys Ile Leu Lys Arg Val Lys Gln Tyr Thr
465 470 475 480
Leu Glu His Ile Met Tyr Leu Gly Lys Leu Arg His Asn Asp Ile Asp
485 490 495
Met Thr Thr Val Asn Thr Asp Asp Phe Ser Arg Leu His Ala Lys Glu
500 505 510
Glu Leu Asp Leu Glu Leu Ile Thr Phe Phe Ala Ser Thr Asn Met Glu
515 520 525
Leu Asn Lys Ile Phe Ser Arg Glu Asn Ile Asn Asn Asp Glu Asn Ile
530 535 540
Asp Phe Phe Gly Gly Asp Arg Glu Lys Asn Tyr Val Leu Asp Lys Lys
545 550 555 560
Ile Leu Asn Ser Lys Ile Lys Ile Ile Arg Asp Leu Asp Phe Ile Asp
565 570 575
Asn Lys Asn Asn Ile Thr Asn Asn Phe Ile Arg Lys Phe Thr Lys Ile
580 585 590
Gly Thr Asn Glu Arg Asn Arg Ile Leu His Ala Ile Ser Lys Glu Arg
595 600 605
Asp Leu Gln Gly Thr Gln Asp Asp Tyr Asn Lys Val Ile Asn Ile Ile
610 615 620
Gln Asn Leu Lys Ile Ser Asp Glu Glu Val Ser Lys Ala Leu Asn Leu
625 630 635 640
Asp Val Val Phe Lys Asp Lys Lys Asn Ile Ile Thr Lys Ile Asn Asp
645 650 655
Ile Lys Ile Ser Glu Glu Asn Asn Asn Asp Ile Lys Tyr Leu Pro Ser
660 665 670
Phe Ser Lys Val Leu Pro Glu Ile Leu Asn Leu Tyr Arg Asn Asn Pro
675 680 685
Lys Asn Glu Pro Phe Asp Thr Ile Glu Thr Glu Lys Ile Val Leu Asn
690 695 700
Ala Leu Ile Tyr Val Asn Lys Glu Leu Tyr Lys Lys Leu Ile Leu Glu
705 710 715 720
Asp Asp Leu Glu Glu Asn Glu Ser Lys Asn Ile Phe Leu Gln Glu Leu
725 730 735
Lys Lys Thr Leu Gly Asn Ile Asp Glu Ile Asp Glu Asn Ile Ile Glu
740 745 750
Asn Tyr Tyr Lys Asn Ala Gln Ile Ser Ala Ser Lys Gly Asn Asn Lys
755 760 765
Ala Ile Lys Lys Tyr Gln Lys Lys Val Ile Glu Cys Tyr Ile Gly Tyr
770 775 780
Leu Arg Lys Asn Tyr Glu Glu Leu Phe Asp Phe Ser Asp Phe Lys Met
785 790 795 800
Asn Ile Gln Glu Ile Lys Lys Gln Ile Lys Asp Ile Asn Asp Asn Lys
805 810 815
Thr Tyr Glu Arg Ile Thr Val Lys Thr Ser Asp Lys Thr Ile Val Ile
820 825 830
Asn Asp Asp Phe Glu Tyr Ile Ile Ser Ile Phe Ala Leu Leu Asn Ser
835 840 845
Asn Ala Val Ile Asn Lys Ile Arg Asn Arg Phe Phe Ala Thr Ser Val
850 855 860
Trp Leu Asn Thr Ser Glu Tyr Gln Asn Ile Ile Asp Ile Leu Asp Glu
865 870 875 880
Ile Met Gln Leu Asn Thr Leu Arg Asn Glu Cys Ile Thr Glu Asn Trp
885 890 895
Asn Leu Asn Leu Glu Glu Phe Ile Gln Lys Met Lys Glu Ile Glu Lys
900 905 910
Asp Phe Asp Asp Phe Lys Ile Gln Thr Lys Lys Glu Ile Phe Asn Asn
915 920 925
Tyr Tyr Glu Asp Ile Lys Asn Asn Ile Leu Thr Glu Phe Lys Asp Asp
930 935 940
Ile Asn Gly Cys Asp Val Leu Glu Lys Lys Leu Glu Lys Ile Val Ile
945 950 955 960
Phe Asp Asp Glu Thr Lys Phe Glu Ile Asp Lys Lys Ser Asn Ile Leu
965 970 975
Gln Asp Glu Gln Arg Lys Leu Ser Asn Ile Asn Lys Lys Asp Leu Lys
980 985 990
Lys Lys Val Asp Gln Tyr Ile Lys Asp Lys Asp Gln Glu Ile Lys Ser
995 1000 1005
Lys Ile Leu Cys Arg Ile Ile Phe Asn Ser Asp Phe Leu Lys Lys
1010 1015 1020
Tyr Lys Lys Glu Ile Asp Asn Leu Ile Glu Asp Met Glu Ser Glu
1025 1030 1035
Asn Glu Asn Lys Phe Gln Glu Ile Tyr Tyr Pro Lys Glu Arg Lys
1040 1045 1050
Asn Glu Leu Tyr Ile Tyr Lys Lys Asn Leu Phe Leu Asn Ile Gly
1055 1060 1065
Asn Pro Asn Phe Asp Lys Ile Tyr Gly Leu Ile Ser Asn Asp Ile
1070 1075 1080
Lys Met Ala Asp Ala Lys Phe Leu Phe Asn Ile Asp Gly Lys Asn
1085 1090 1095
Ile Arg Lys Asn Lys Ile Ser Glu Ile Asp Ala Ile Leu Lys Asn
1100 1105 1110
Leu Asn Asp Lys Leu Asn Gly Tyr Ser Lys Glu Tyr Lys Glu Lys
1115 1120 1125
Tyr Ile Lys Lys Leu Lys Glu Asn Asp Asp Phe Phe Ala Lys Asn
1130 1135 1140
Ile Gln Asn Lys Asn Tyr Lys Ser Phe Glu Lys Asp Tyr Asn Arg
1145 1150 1155
Val Ser Glu Tyr Lys Lys Ile Arg Asp Leu Val Glu Phe Asn Tyr
1160 1165 1170
Leu Asn Lys Ile Glu Ser Tyr Leu Ile Asp Ile Asn Trp Lys Leu
1175 1180 1185
Ala Ile Gln Met Ala Arg Phe Glu Arg Asp Met His Tyr Ile Val
1190 1195 1200
Asn Gly Leu Arg Glu Leu Gly Ile Ile Lys Leu Ser Gly Tyr Asn
1205 1210 1215
Thr Gly Ile Ser Arg Ala Tyr Pro Lys Arg Asn Gly Ser Asp Gly
1220 1225 1230
Phe Tyr Thr Thr Thr Ala Tyr Tyr Lys Phe Phe Asp Glu Glu Ser
1235 1240 1245
Tyr Lys Lys Phe Glu Lys Ile Cys Tyr Gly Phe Gly Ile Asp Leu
1250 1255 1260
Ser Glu Asn Ser Glu Ile Asn Lys Pro Glu Asn Glu Ser Ile Arg
1265 1270 1275
Asn Tyr Ile Ser His Phe Tyr Ile Val Arg Asn Pro Phe Ala Asp
1280 1285 1290
Tyr Ser Ile Ala Glu Gln Ile Asp Arg Val Ser Asn Leu Leu Ser
1295 1300 1305
Tyr Ser Thr Arg Tyr Asn Asn Ser Thr Tyr Ala Ser Val Phe Glu
1310 1315 1320
Val Phe Lys Lys Asp Val Asn Leu Asp Tyr Asp Glu Leu Lys Lys
1325 1330 1335
Lys Phe Lys Leu Ile Gly Asn Asn Asp Ile Leu Glu Arg Leu Met
1340 1345 1350
Lys Pro Lys Lys Val Ser Val Leu Glu Leu Glu Ser Tyr Asn Ser
1355 1360 1365
Asp Tyr Ile Lys Asn Leu Ile Ile Glu Leu Leu Thr Lys Ile Glu
1370 1375 1380
Asn Thr Asn Asp Thr Leu Lys Arg Pro Ala Ala Thr Lys Lys Ala
1385 1390 1395
Gly Gln Ala Lys Lys Lys Lys Gly Ser Tyr Pro Tyr Asp Val Pro
1400 1405 1410
Asp Tyr Ala Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Tyr Pro Tyr
1415 1420 1425
Asp Val Pro Asp Tyr Ala
1430
<210> 72
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 72
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 73
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 73
ccaccccaau aucgaagggg acuaaaacuu ucuagaggau ccccggguac cgagcu 56
<210> 74
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 74
ccaccccaau aucgaagggg acuaaaacag uaauccauau uucuagagga uccccg 56
<210> 75
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 75
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 76
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 76
ccaccccaau aucgaagggg acuaaaacca ugccugcagg ucgaguagau ugcugu 56
<210> 77
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 77
ccaccccaau aucgaagggg acuaaaacgc augccugcag gucgaguaga uugcug 56
<210> 78
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 78
ccaccccaau aucgaagggg acuaaaacaa gcuugcaugc cugcaggucg aguaga 56
<210> 79
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 79
ccaccccaau aucgaagggg acuaaaaccg ccaagcuugc augccugcag gucgag 56
<210> 80
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 80
ccaccccaau aucgaagggg acuaaaacga uuacgccaag cuugcaugcc ugcagg 56
<210> 81
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 81
ccaccccaau aucgaagggg acuaaaacug auuacgccaa gcuugcaugc cugcag 56
<210> 82
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 82
ccaccccaau aucgaagggg acuaaaacau gaccaugauu acgccaagcu ugcaug 56
<210> 83
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 83
ccaccccaau aucgaagggg acuaaaacua ugaccaugau uacgccaagc uugcau 56
<210> 84
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 84
ccaccccaau aucgaagggg acuaaaacag cuaugaccau gauuacgcca agcuug 56
<210> 85
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 85
ccaccccaau aucgaagggg acuaaaacga aacagcuaug accaugauua cgccaa 56
<210> 86
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 86
ccaccccaau aucgaagggg acuaaaacac aggaaacagc uaugaccaug auuacg 56
<210> 87
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 87
ccaccccaau aucgaagggg acuaaaacaa cacaggaaac agcuaugacc augauu 56
<210> 88
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 88
ccaccccaau aucgaagggg acuaaaacaa acacaggaaa cagcuaugac caugau 56
<210> 89
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 89
ccaccccaau aucgaagggg acuaaaacau aaacacagga aacagcuaug accaug 56
<210> 90
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 90
ccaccccaau aucgaagggg acuaaaacgg auaaacacag gaaacagcua ugacca 56
<210> 91
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 91
ccaccccaau aucgaagggg acuaaaacag cggauaaaca caggaaacag cuauga 56
<210> 92
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 92
ccaccccaau aucgaagggg acuaaaacga gcggauaaac acaggaaaca gcuaug 56
<210> 93
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 93
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 94
<211> 54
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 94
accccaauau cgaaggggac uaaaacuaga uugcuguucu accaaguaau ccau 54
<210> 95
<211> 52
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 95
cccaauaucg aaggggacua aaacuagauu gcuguucuac caaguaaucc au 52
<210> 96
<211> 50
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 96
caauaucgaa ggggacuaaa acuagauugc uguucuacca aguaauccau 50
<210> 97
<211> 48
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 97
auaucgaagg ggacuaaaac uagauugcug uucuaccaag uaauccau 48
<210> 98
<211> 47
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 98
uaucgaaggg gacuaaaacu agauugcugu ucuaccaagu aauccau 47
<210> 99
<211> 46
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 99
aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 46
<210> 100
<211> 52
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 100
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua au 52
<210> 101
<211> 51
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 101
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua a 51
<210> 102
<211> 50
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 102
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua 50
<210> 103
<211> 49
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 103
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagu 49
<210> 104
<211> 48
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 104
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaag 48
<210> 105
<211> 47
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 105
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaa 47
<210> 106
<211> 46
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 106
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuacca 46
<210> 107
<211> 45
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 107
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuacc 45
<210> 108
<211> 44
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 108
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuac 44
<210> 109
<211> 40
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 109
ccaccccaau aucgaagggg acuaaaacua gauugcuguu 40
<210> 110
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 110
ccacccgaau aucgaacggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 111
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 111
ccaccgcaau aucgaagcgg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 112
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 112
ccacgccaau aucgaaggcg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 113
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 113
ccagcccaau aucgaagggc acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 114
<211> 57
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 114
ccagggggaa uaucgaaccc cacuaaaacu agauugcugu ucuaccaagu aauccau 57
<210> 115
<211> 57
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 115
ccaccaccaa uaucgaaggg gacuaaaacu agauugcugu ucuaccaagu aauccau 57
<210> 116
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 116
ccaacccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 117
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 117
ccacccaaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 118
<211> 58
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 118
ccacccccaa uaucgaaggg ggacuaaaac uagauugcug uucuaccaag uaauccau 58
<210> 119
<211> 55
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 119
ccaccccaua ucgaagggga cuaaaacuag auugcuguuc uaccaaguaa uccau 55
<210> 120
<211> 53
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 120
ccaccccauc gaaggggacu aaaacuagau ugcuguucua ccaaguaauc cau 53
<210> 121
<211> 49
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 121
ccaccccaag gggacuaaaa cuagauugcu guucuaccaa guaauccau 49
<210> 122
<211> 57
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 122
ccaccccaaa uaucgaaggg gacuaaaacu agauugcugu ucuaccaagu aauccau 57
<210> 123
<211> 59
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 123
ccaccccaaa aauaucgaag gggacuaaaa cuagauugcu guucuaccaa guaauccau 59
<210> 124
<211> 63
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 124
ccaccccaaa aaaaaauauc gaaggggacu aaaacuagau ugcuguucua ccaaguaauc 60
cau 63
<210> 125
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 125
ccaccccgau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 126
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 126
ccaccccaaa aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 127
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 127
ccaccccaau auccaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 128
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 128
ccaccccaau aucgaagggg acuaaaacaa gauugcuguu cuaccaagua auccau 56
<210> 129
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 129
ccaccccaau aucgaagggg acuaaaacua gaaugcuguu cuaccaagua auccau 56
<210> 130
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 130
ccaccccaau aucgaagggg acuaaaacua gauugcaguu cuaccaagua auccau 56
<210> 131
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 131
ccaccccaau aucgaagggg acuaaaacua gauugcuguu guaccaagua auccau 56
<210> 132
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 132
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuacgaagua auccau 56
<210> 133
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 133
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagaa auccau 56
<210> 134
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 134
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua augcau 56
<210> 135
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 135
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccaa 56
<210> 136
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 136
ccaccccaau aucgaagggg acuaaaacau gauugcuguu cuaccaagua auccau 56
<210> 137
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 137
ccaccccaau aucgaagggg acuaaaacua gauaccuguu cuaccaagua auccau 56
<210> 138
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 138
ccaccccaau aucgaagggg acuaaaacua gauugcugaa cuaccaagua auccau 56
<210> 139
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 139
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaggaagua auccau 56
<210> 140
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 140
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagau auccau 56
<210> 141
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 141
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua aucguu 56
<210> 142
<211> 173
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 142
ggccagugaa uucgagcucg guacccgggg auccucuaga aauauggauu acuugguaga 60
acagcaaucu acucgaccug caggcaugca agcuuggcgu aaucaugguc auagcuguuu 120
ccuguguuua uccgcucaca auuccacaca acauacgagc cggaagcaua aag 173
<210> 143
<211> 152
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 143
aauauggauu acuugguaga acagcaaucu acaaaaaaaa aaaaaaaaaa agaaaaaaaa 60
aaaaaaaaaa agaaaaaaaa aaaaaaaaaa agaaaaaaaa aaaaaaaaaa agaaaaaaaa 120
aaaaaaaaaa agaaaaaaaa aaaaaaaaaa ag 152
<210> 144
<211> 152
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 144
aauauggauu acuugguaga acagcaaucu acuuuuuuuu uuuuuuuuuu ucuuuuuuuu 60
uuuuuuuuuu ucuuuuuuuu uuuuuuuuuu ucuuuuuuuu uuuuuuuuuu ucuuuuuuuu 120
uuuuuuuuuu ucuuuuuuuu uuuuuuuuuu uc 152
<210> 145
<211> 176
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 145
ggguaggugu uccacagggu agccagcagc auccugcgau gcaaauaugg auuacuuggu 60
agaacagcaa ucuaauccgg aacauaaugg ugcagggcgc ugacuuccgc guuuccagac 120
uuuacgaaac acggaaaccg aagaccauuc auguuguugc ugccggaagc auaaag 176
<210> 146
<211> 176
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 146
gggccccucc guucgcguuu acgcggacgg ugagacugaa gauaauaugg auuacuuggu 60
agaacagcaa ucuaaacuca uucucuuuaa aauaucguuc gaacuggacu cccggucguu 120
uuaacucgac uggggccaaa acgaaacagu ggcacuaccc cgccggaagc auaaag 176
<210> 147
<211> 173
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 147
ggccagugaa uucgagcucg guacccgggg auccucuaga aauauggauu acuugguaga 60
acagcaaucu agucgaccug caggcaugca agcuuggcgu aaucaugguc auagcuguuu 120
ccuguguuua uccgcucaca auuccacaca acauacgagc cggaagcaua aag 173
<210> 148
<211> 173
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 148
ggccagugaa uucgagcucg guacccgggg auccucuaga aauauggauu acuugguaga 60
acagcaaucu aaucgaccug caggcaugca agcuuggcgu aaucaugguc auagcuguuu 120
ccuguguuua uccgcucaca auuccacaca acauacgagc cggaagcaua aag 173
<210> 149
<211> 173
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 149
ggccagugaa uucgagcucg guacccgggg auccucuaga aauauggauu acuugguaga 60
acagcaaucu auucgaccug caggcaugca agcuuggcgu aaucaugguc auagcuguuu 120
ccuguguuua uccgcucaca auuccacaca acauacgagc cggaagcaua aag 173
<210> 150
<211> 175
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 150
accgaucguc guuguuuggg caaugcacgu ucuccaacgg ugcuccuaug gggcacaagu 60
ugcaggaugc agcgccuuac aagaaguucg cugaacaagc aaccguuacc ccccgcgcuc 120
ugagagcggc ucuauugguc cgagaccaau gugcgccgug gaucagacac gcggu 175
<210> 151
<211> 175
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 151
acuguuggug guguagagcu uccuguagcc gcauggcguu cguacuuaaa uauggaacua 60
accauuccaa uuuucgcuac gaauuccgac ugcgagcuua uuguuaaggc aaugcaaggu 120
cuccuaaaag auggaaaccc gauucccuca gcaaucgcag caaacuccgg caucu 175
<210> 152
<211> 175
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 152
gguaacaugc ucgagggccu uacggccccc gugggaugcu ccuacauguc aggaacaguu 60
acugacguaa uaacggguga guccaucaua agcguugacg cucccuacgg guggacugug 120
gagagacagg gcacugcuaa ggcccaaauc ucagccaugc aucgaggggu acaau 175
<210> 153
<211> 175
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 153
uucguaaaac guucgugucc gggcucuuuc gcgagagcug cggcgcgcac uuuuaccgug 60
gugucgaugu caaaccguuu uacaucaaga aaccuguuga caaucucuuc gcccugaugc 120
ugauauuaaa ucggcuacgg gguuggggag uugucggagg uaugucagau ccacg 175
<210> 154
<211> 70
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 154
auaggccagu gaauucgagc ucgaauaugg auuacuuggu agaacagcaa ucuacgccgg 60
aagcauaaag 70
<210> 155
<211> 70
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 155
cuuuaugcuu ccggcguaga uugcuguucu accaaguaau ccauauucga gcucgaauuc 60
acuggccuau 70
<210> 156
<211> 198
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 156
ctttatgctt ccggctcgta tgttgtgtgg aattgtgagc ggataaacac aggaaacagc 60
tatgaccatg attacgccaa gcttgcatgc ctgcaggtcg agaatatgga ttacttggta 120
gaacagcaat ctatctagag gatccccggg taccgagctc gaattcactg gccccctata 180
gtgagtcgta ttaatttc 198
<210> 157
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 157
gaaguuugca gcuggauacg acagacgg 28
<210> 158
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 158
ugucuggaag uuugcagcug gauacgac 28
<210> 159
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 159
agcuggauac gacagacggc caucuaac 28
<210> 160
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 160
uacgucgcga uauguugcac guugucug 28
<210> 161
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 161
uacggacgac cuucaccuuc accuucgauu u 31
<210> 162
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 162
ucguacggac gaccuucacc uucaccuucg a 31
<210> 163
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 163
cggucugggu accuucguac ggacgaccuu c 31
<210> 164
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 164
gcggucuggg uaccuucgua cggacgaccu u 31
<210> 165
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 165
agcggucugg guaccuucgu acggacgacc u 31
<210> 166
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 166
aguucauaac acguucccau uugaaaccuu c 31
<210> 167
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 167
uuaacuuugu agaugaacuc accgucuugc a 31
<210> 168
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 168
uuuaacuuug uagaugaacu caccgucuug c 31
<210> 169
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 169
guuuaacuuu guagaugaac ucaccgucuu g 31
<210> 170
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 170
aaguuugcag cuggauacga cagacggc 28
<210> 171
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 171
acaggauguc ccaagcgaac ggcagcgg 28
<210> 172
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 172
gcuuguucag cgaacuucuu guaaggcg 28
<210> 173
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 173
uaagcucgca gucggaauuc guagcgaa 28
<210> 174
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 174
cgucaacgcu uaugauggac ucacccgu 28
<210> 175
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 175
ucaacagguu ucuugaugua aaacgguu 28
<210> 176
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 176
aaguuugcag cuggauacga cagacggc 28
<210> 177
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 177
uuugcagcug gauacgacag acggccau 28
<210> 178
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 178
cagcuggaua cgacagacgg ccaucuaa 28
<210> 179
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 179
guugucugga aguuugcagc uggauacg 28
<210> 180
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 180
gcgauauguu gcacguuguc uggaaguu 28
<210> 181
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 181
acguugucug gaaguuugca gcuggaua 28
<210> 182
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 182
auguugcacg uugucuggaa guuugcag 28
<210> 183
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 183
ugcagcugga uacgacagac ggccaucu 28
<210> 184
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 184
cuggaaguuu gcagcuggau acgacaga 28
<210> 185
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 185
aguuugcagc uggauacgac agacggcc 28
<210> 186
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 186
gcuggauacg acagacggcc aucuaacu 28
<210> 187
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 187
guuugcagcu ggauacgaca gacggcca 28
<210> 188
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 188
uaguuugcag cuggauacga cagacggc 28
<210> 189
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 189
aaguaugcag cuggauacga cagacggc 28
<210> 190
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 190
aaguuugcug cuggauacga cagacggc 28
<210> 191
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 191
aaguuugcag cucgauacga cagacggc 28
<210> 192
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 192
aaguuugcag cuggauucga cagacggc 28
<210> 193
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 193
aaguuugcag cuggauacga gagacggc 28
<210> 194
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 194
aaguuugcag cuggauacga cagagggc 28
<210> 195
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 195
uuguuugcag cuggauacga cagacggc 28
<210> 196
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 196
aaguuaccag cuggauacga cagacggc 28
<210> 197
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 197
aaguuugcag gaggauacga cagacggc 28
<210> 198
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 198
aaguuugcag cuggaaucga cagacggc 28
<210> 199
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 199
aaguuugcag cuggauacga gugacggc 28
<210> 200
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(6)
<223> /note="Description of Artificial Sequence: Synthetic 6xHis tag"
<400> 200
His His His His His His
1 5
<210> 201
<211> 5
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(5)
<223> /note="Description of Unknown: 'GGDEF' family motif peptide"
<400> 201
Gly Gly Asp Glu Phe
1 5
<210> 202
<211> 18
<212> PRT
<213> Alicyclobacillus acidoterrestris
<400> 202
Leu Arg Val Met Ser Val Asp Leu Gly Leu Arg Thr Ser Ala Ser Ile
1 5 10 15
Ser Val
<210> 203
<211> 22
<212> PRT
<213> Alicyclobacillus acidoterrestris
<400> 203
Gln Arg Thr Leu Arg Gln Leu Arg Thr Gln Leu Ala Tyr Leu Arg Leu
1 5 10 15
Leu Val Arg Cys Gly Ser
20
<210> 204
<211> 12
<212> PRT
<213> Alicyclobacillus acidoterrestris
<400> 204
Cys Gln Leu Ile Leu Leu Glu Glu Leu Ser Glu Tyr
1 5 10
<210> 205
<211> 16
<212> PRT
<213> Alicyclobacillus acidoterrestris
<400> 205
His Gln Ile His Ala Asp Leu Asn Ala Ala Gln Asn Leu Gln Gln Arg
1 5 10 15
<210> 206
<211> 18
<212> PRT
<213> Alicyclobacillus contaminans
<400> 206
Val Arg Val Met Ser Val Asp Leu Gly Val Arg Tyr Gly Ala Ala Ile
1 5 10 15
Ser Val
<210> 207
<211> 22
<212> PRT
<213> Alicyclobacillus contaminans
<400> 207
Lys Gln Ala Leu Ala Ala Ile Arg Ala Glu Met Ser Ile Leu Arg Lys
1 5 10 15
Trp Leu Arg Val Ser Gln
20
<210> 208
<211> 12
<212> PRT
<213> Alicyclobacillus contaminans
<400> 208
Cys Asp Leu Ile Leu Phe Glu Asp Leu Ser Arg Tyr
1 5 10
<210> 209
<211> 16
<212> PRT
<213> Alicyclobacillus contaminans
<400> 209
Lys Cys Val His Ala Asp Ile Asn Ala Ala His Asn Leu Gln Arg Arg
1 5 10 15
<210> 210
<211> 18
<212> PRT
<213> Desulfovibrio inopinatus
<400> 210
Leu Arg Val Leu Ser Val Asp Leu Gly Met Arg Thr Phe Ala Ser Cys
1 5 10 15
Ser Val
<210> 211
<211> 22
<212> PRT
<213> Desulfovibrio inopinatus
<400> 211
Arg Ala Glu Ile Tyr Ala Leu Lys Arg Asp Ile Gln Arg Leu Lys Ser
1 5 10 15
Leu Leu Arg Leu Gly Glu
20
<210> 212
<211> 12
<212> PRT
<213> Desulfovibrio inopinatus
<400> 212
Cys Gln Leu Ile Leu Phe Glu Asp Leu Ala Arg Tyr
1 5 10
<210> 213
<211> 16
<212> PRT
<213> Desulfovibrio inopinatus
<400> 213
Cys Val Ile His Ala Asp Met Asn Ala Ala Gln Asn Leu Gln Arg Arg
1 5 10 15
<210> 214
<211> 18
<212> PRT
<213> Desulfonatronum thiodismutans
<400> 214
Leu Arg Val Leu Ser Val Asp Leu Gly Val Arg Ser Phe Ala Ala Cys
1 5 10 15
Ser Val
<210> 215
<211> 22
<212> PRT
<213> Desulfonatronum thiodismutans
<400> 215
Met Glu Glu Leu Arg Ser Leu Asn Gly Asp Ile Arg Arg Leu Lys Ala
1 5 10 15
Ile Leu Arg Leu Ser Val
20
<210> 216
<211> 12
<212> PRT
<213> Desulfonatronum thiodismutans
<400> 216
Cys Arg Leu Ile Leu Phe Glu Asp Leu Ala Arg Tyr
1 5 10
<210> 217
<211> 16
<212> PRT
<213> Desulfonatronum thiodismutans
<400> 217
His Val Ile His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln Arg Arg
1 5 10 15
<210> 218
<211> 18
<212> PRT
<213> Tuberibacillus calidus
<400> 218
Leu Arg Val Met Ser Val Asp Leu Gly Gln Arg Gln Ala Ala Ala Ile
1 5 10 15
Ser Ile
<210> 219
<211> 22
<212> PRT
<213> Tuberibacillus calidus
<400> 219
Asp Gln Ala Ile Arg Asp Leu Ser Arg Lys Leu Lys Phe Leu Lys Asn
1 5 10 15
Val Leu Asn Met Gln Lys
20
<210> 220
<211> 12
<212> PRT
<213> Tuberibacillus calidus
<400> 220
Cys Gln Leu Val Leu Phe Glu Asp Leu Ser Arg Tyr
1 5 10
<210> 221
<211> 16
<212> PRT
<213> Tuberibacillus calidus
<400> 221
Val Ile Thr His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln Lys Arg
1 5 10 15
<210> 222
<211> 18
<212> PRT
<213> Bacillus thermoamylovorans
<400> 222
Leu Arg Val Met Ser Ile Asp Leu Gly Gln Arg Gln Ala Ala Ala Ala
1 5 10 15
Ser Ile
<210> 223
<211> 22
<212> PRT
<213> Bacillus thermoamylovorans
<400> 223
Glu Asp Asn Leu Lys Leu Met Asn Gln Lys Leu Asn Phe Leu Arg Asn
1 5 10 15
Val Leu His Phe Gln Gln
20
<210> 224
<211> 12
<212> PRT
<213> Bacillus thermoamylovorans
<400> 224
Cys Gln Ile Ile Leu Phe Glu Asp Leu Ser Asn Tyr
1 5 10
<210> 225
<211> 16
<212> PRT
<213> Bacillus thermoamylovorans
<400> 225
Val Thr Thr His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln Lys Arg
1 5 10 15
<210> 226
<211> 18
<212> PRT
<213> Bacillus sp.
<400> 226
Phe Arg Val Met Ser Ile Asp Leu Gly Leu Arg Ala Ala Ala Ala Thr
1 5 10 15
Ser Ile
<210> 227
<211> 22
<212> PRT
<213> Bacillus sp.
<400> 227
Phe Gln Leu His Gln Arg Val Lys Phe Gln Ile Arg Val Leu Ala Gln
1 5 10 15
Ile Met Arg Met Ala Asn
20
<210> 228
<211> 12
<212> PRT
<213> Bacillus sp.
<400> 228
Cys Gln Val Ile Leu Phe Glu Asn Leu Ser Gln Tyr
1 5 10
<210> 229
<211> 16
<212> PRT
<213> Bacillus sp.
<400> 229
Val Phe Leu Gln Ala Asp Ile Asn Ala Ala His Asn Leu Gln Lys Arg
1 5 10 15
<210> 230
<211> 18
<212> PRT
<213> Methylobacterium nodulans
<400> 230
Leu Arg Val Leu Ser Ile Asp Leu Gly Val Arg Ser Phe Ala Thr Cys
1 5 10 15
Ser Val
<210> 231
<211> 22
<212> PRT
<213> Methylobacterium nodulans
<400> 231
Asp Ala Glu Leu Arg Gln Leu Arg Gly Gly Leu Asn Arg His Arg Gln
1 5 10 15
Leu Leu Arg Ala Ala Thr
20
<210> 232
<211> 12
<212> PRT
<213> Methylobacterium nodulans
<400> 232
Cys His Val Ile Leu Phe Glu Asp Leu Ser Arg Tyr
1 5 10
<210> 233
<211> 16
<212> PRT
<213> Methylobacterium nodulans
<400> 233
Ser Arg Ile His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln Arg Arg
1 5 10 15
<210> 234
<211> 18
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Candidatus Methanomethylophilus
alvus sequence"
<400> 234
Leu Lys Ile Ile Gly Ile Asp Arg Gly Glu Arg Asn Leu Ile Tyr Val
1 5 10 15
Thr Met
<210> 235
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Candidatus Methanomethylophilus
alvus sequence"
<400> 235
Arg Lys Ala Leu Asp Val Arg Glu Tyr Asp Asn Lys Glu Ala Arg Arg
1 5 10 15
Asn Trp Thr Lys Val Glu
20
<210> 236
<211> 13
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(13)
<223> /note="Description of Unknown: Candidatus Methanomethylophilus
alvus sequence"
<400> 236
Asn Ala Ile Ile Val Met Glu Asp Leu Asn His Gly Phe
1 5 10
<210> 237
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Candidatus Methanomethylophilus
alvus sequence"
<400> 237
Leu Pro Gln Asp Ser Asp Ala Asn Gly Ala Tyr Asn Ile Ala Leu Lys
1 5 10 15
<210> 238
<211> 18
<212> PRT
<213> Synergistes jonesii
<400> 238
Val Asn Ile Ile Gly Ile Asp Arg Gly Glu Arg Asn Leu Val Tyr Val
1 5 10 15
Ser Leu
<210> 239
<211> 22
<212> PRT
<213> Synergistes jonesii
<400> 239
His Ala Lys Leu Asn Gln Lys Glu Lys Glu Arg Asp Thr Ala Arg Lys
1 5 10 15
Ser Trp Lys Thr Ile Gly
20
<210> 240
<211> 13
<212> PRT
<213> Synergistes jonesii
<400> 240
Asn Ala Val Ile Val Met Glu Asp Leu Asn Ile Gly Phe
1 5 10
<210> 241
<211> 16
<212> PRT
<213> Synergistes jonesii
<400> 241
Leu Pro Ile Asp Ala Asp Ala Asn Gly Ala Tyr His Ile Ala Leu Lys
1 5 10 15
<210> 242
<211> 18
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 242
Pro Tyr Val Ile Gly Ile Asp Arg Gly Glu Arg Asn Leu Leu Tyr Ile
1 5 10 15
Val Val
<210> 243
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 243
His Ser Leu Leu Asp Lys Lys Glu Lys Glu Arg Phe Glu Ala Arg Gln
1 5 10 15
Asn Trp Thr Ser Ile Glu
20
<210> 244
<211> 13
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(13)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 244
Asp Ala Val Ile Ala Leu Glu Asp Leu Asn Ser Gly Phe
1 5 10
<210> 245
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 245
Leu Pro Lys Asn Ala Asp Ala Asn Gly Ala Tyr Asn Ile Ala Arg Lys
1 5 10 15
<210> 246
<211> 18
<212> PRT
<213> Francisella tularensis
<400> 246
Val His Ile Leu Ser Ile Asp Arg Gly Glu Arg His Leu Ala Tyr Tyr
1 5 10 15
Thr Leu
<210> 247
<211> 22
<212> PRT
<213> Francisella tularensis
<400> 247
His Asp Lys Leu Ala Ala Ile Glu Lys Asp Arg Asp Ser Ala Arg Lys
1 5 10 15
Asp Trp Lys Lys Ile Asn
20
<210> 248
<211> 13
<212> PRT
<213> Francisella tularensis
<400> 248
Asn Ala Ile Val Val Phe Glu Asp Leu Asn Phe Gly Phe
1 5 10
<210> 249
<211> 16
<212> PRT
<213> Francisella tularensis
<400> 249
Met Pro Gln Asp Ala Asp Ala Asn Gly Ala Tyr His Ile Gly Leu Lys
1 5 10 15
<210> 250
<211> 18
<212> PRT
<213> Moraxella caprae
<400> 250
Val Asn Val Ile Gly Ile Asp Arg Gly Glu Arg His Leu Leu Tyr Leu
1 5 10 15
Thr Val
<210> 251
<211> 22
<212> PRT
<213> Moraxella caprae
<400> 251
His Lys Ile Leu Asp Lys Arg Glu Ile Glu Arg Leu Asn Ala Arg Val
1 5 10 15
Gly Trp Gly Glu Ile Glu
20
<210> 252
<211> 13
<212> PRT
<213> Moraxella caprae
<400> 252
Asn Ala Ile Val Val Leu Glu Asp Leu Asn Phe Gly Phe
1 5 10
<210> 253
<211> 16
<212> PRT
<213> Moraxella caprae
<400> 253
Gln Pro Gln Asn Ala Asp Ala Asn Gly Ala Tyr His Ile Ala Leu Lys
1 5 10 15
<210> 254
<211> 18
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 254
Met His Ile Ile Gly Ile Asp Arg Gly Glu Arg Asn Leu Ile Tyr Leu
1 5 10 15
Cys Met
<210> 255
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 255
His Gln Leu Leu Lys Thr Arg Glu Asp Glu Asn Lys Ser Ala Arg Gln
1 5 10 15
Ser Trp Gln Thr Ile His
20
<210> 256
<211> 13
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(13)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 256
Asn Ala Ile Val Val Leu Glu Asp Leu Asn Phe Gly Phe
1 5 10
<210> 257
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(6)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 257
Met Pro Leu Asp Ala Asp Ala Asn Gly Ala Tyr Asn Ile Ala Arg Lys
1 5 10 15
<210> 258
<211> 18
<212> PRT
<213> Prevotella albensis
<400> 258
Thr His Ile Ile Gly Ile Asp Arg Gly Glu Arg His Leu Leu Tyr Leu
1 5 10 15
Ser Leu
<210> 259
<211> 22
<212> PRT
<213> Prevotella albensis
<400> 259
His Asn Leu Leu Glu Lys Arg Glu Lys Glu Arg Thr Glu Ala Arg His
1 5 10 15
Ser Trp Ser Ser Ile Glu
20
<210> 260
<211> 13
<212> PRT
<213> Prevotella albensis
<400> 260
Asn Ala Ile Val Val Leu Glu Asp Leu Asn Gly Gly Phe
1 5 10
<210> 261
<211> 16
<212> PRT
<213> Prevotella albensis
<400> 261
Phe Pro Glu Asn Ala Asp Ala Asn Gly Ala Tyr Asn Ile Ala Arg Lys
1 5 10 15
<210> 262
<211> 18
<212> PRT
<213> Smithella sp.
<400> 262
Ile Asn Ile Ile Gly Ile Asp Arg Gly Glu Arg His Leu Leu Tyr Tyr
1 5 10 15
Ala Leu
<210> 263
<211> 22
<212> PRT
<213> Smithella sp.
<400> 263
His Asn Leu Leu Asp Lys Lys Glu Gly Asp Arg Ala Thr Ala Arg Gln
1 5 10 15
Glu Trp Gly Val Ile Glu
20
<210> 264
<211> 13
<212> PRT
<213> Smithella sp.
<400> 264
Asn Ala Ile Ile Val Met Glu Asp Leu Asn Phe Gly Phe
1 5 10
<210> 265
<211> 16
<212> PRT
<213> Smithella sp.
<400> 265
Met Pro Lys Asn Ala Asp Ala Asn Gly Ala Tyr His Ile Ala Leu Lys
1 5 10 15
<210> 266
<211> 18
<212> PRT
<213> Porphyromonas crevioricanis
<400> 266
Met His Val Ile Gly Ile Asp Arg Gly Glu Arg Asn Leu Leu Tyr Ile
1 5 10 15
Cys Val
<210> 267
<211> 22
<212> PRT
<213> Porphyromonas crevioricanis
<400> 267
His Asp Leu Leu Glu Ser Arg Asp Lys Asp Arg Gln Gln Glu Arg Arg
1 5 10 15
Asn Trp Gln Thr Ile Glu
20
<210> 268
<211> 13
<212> PRT
<213> Porphyromonas crevioricanis
<400> 268
Lys Ala Val Val Ala Leu Glu Asp Leu Asn Met Gly Phe
1 5 10
<210> 269
<211> 16
<212> PRT
<213> Porphyromonas crevioricanis
<400> 269
Leu Pro Lys Asp Ala Asp Ala Asn Gly Ala Tyr Asn Ile Ala Leu Lys
1 5 10 15
<210> 270
<211> 18
<212> PRT
<213> Sulfolobus solfataricus
<400> 270
Gly Lys Val Val Ala Ile Asp Val Gly Val Glu Lys Leu Leu Ile Thr
1 5 10 15
Ser Asp
<210> 271
<211> 23
<212> PRT
<213> Sulfolobus solfataricus
<400> 271
Val Lys His Ile His Arg Glu Leu Ser Arg Lys Lys Phe Leu Ser Asn
1 5 10 15
Asn Trp Phe Lys Ala Lys Val
20
<210> 272
<211> 13
<212> PRT
<213> Sulfolobus solfataricus
<400> 272
Tyr Asp Val Val Val Met Glu Gly Ile His Ala Lys Gln
1 5 10
<210> 273
<211> 16
<212> PRT
<213> Sulfolobus solfataricus
<400> 273
Trp Ile Ala Asp Arg Asp Tyr Asn Ala Ser Leu Asn Ile Leu Arg Gly
1 5 10 15
<210> 274
<211> 18
<212> PRT
<213> Nostoc sp.
<400> 274
Leu Lys Thr Ile Gly Leu Asp Val Gly Leu Asn His Phe Leu Thr Asp
1 5 10 15
Ser Glu
<210> 275
<211> 23
<212> PRT
<213> Nostoc sp.
<400> 275
Leu Lys Arg Leu Gln Arg Arg Leu Ser Lys Thr Lys Lys Gly Ser Asn
1 5 10 15
Asn Arg Val Lys Ala Arg Asn
20
<210> 276
<211> 13
<212> PRT
<213> Nostoc sp.
<400> 276
Ser Asp Leu Val Ala Tyr Glu Asp Leu Gln Val Arg Asn
1 5 10
<210> 277
<211> 16
<212> PRT
<213> Nostoc sp.
<400> 277
His Ile Gln Asp Arg Asp Trp Asn Ala Ala Arg Asn Ile Leu Glu Leu
1 5 10 15
<210> 278
<211> 18
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Candidatus Nitrososphaera
gargensis sequence"
<400> 278
Ala Lys Pro Val Gly Ile Asp Val Gly Ile Ala Lys Phe Cys His His
1 5 10 15
Ser Asp
<210> 279
<211> 23
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(23)
<223> /note="Description of Unknown: Candidatus Nitrososphaera
gargensis sequence"
<400> 279
Leu Arg Arg Ala His Arg Arg Val Ser Arg Arg Gln Ile Gly Ser Asn
1 5 10 15
Asn Arg Lys Lys Ala Lys Arg
20
<210> 280
<211> 13
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(13)
<223> /note="Description of Unknown: Candidatus Nitrososphaera
gargensis sequence"
<400> 280
Tyr Asp Leu Ile Phe Leu Glu Arg Leu Arg Val Met Asn
1 5 10
<210> 281
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Candidatus Nitrososphaera
gargensis sequence"
<400> 281
Ala Ile Leu Asp Arg Asp Tyr Asn Ser Ala Ile Asn Ile Leu Lys Arg
1 5 10 15
<210> 282
<211> 18
<212> PRT
<213> Helicobacter pylori
<400> 282
Lys Lys Ala Val Gly Leu Asp Met Gly Leu Arg Thr Leu Ile Val Thr
1 5 10 15
Ser Asp
<210> 283
<211> 23
<212> PRT
<213> Helicobacter pylori
<400> 283
Leu Thr Lys Ala Gln Arg Arg Leu Ser Lys Lys Val Lys Asp Ser Asn
1 5 10 15
Asn Arg Lys Lys Gln Ala Lys
20
<210> 284
<211> 13
<212> PRT
<213> Helicobacter pylori
<400> 284
Tyr Asp Leu Ile Gly Val Glu Thr Leu Asn Val Lys Ala
1 5 10
<210> 285
<211> 16
<212> PRT
<213> Helicobacter pylori
<400> 285
Thr Thr His His Arg Asp Tyr Asn Ala Ser Val Asn Ile Arg Asn Tyr
1 5 10 15
<210> 286
<211> 18
<212> PRT
<213> Flexibacter litoralis
<400> 286
Asn Gln Ala Val Gly Ile Asp Met Gly Ile Thr Phe Phe Cys Ile Asp
1 5 10 15
Ser Asn
<210> 287
<211> 23
<212> PRT
<213> Flexibacter litoralis
<400> 287
Leu Arg Ile Ala Asn Arg Ser Leu Ser Arg Lys Lys Lys Phe Ser Asn
1 5 10 15
Gly Trp Tyr Lys Lys Lys Val
20
<210> 288
<211> 13
<212> PRT
<213> Flexibacter litoralis
<400> 288
Asn Ser Leu Val Val Val Glu Asp Leu Lys Val Lys Asn
1 5 10
<210> 289
<211> 16
<212> PRT
<213> Flexibacter litoralis
<400> 289
His Glu Thr Asn Ala Asp Glu Asn Ala Ser Lys Asn Ile Leu Ser Glu
1 5 10 15
<210> 290
<211> 18
<212> PRT
<213> Escherichia coli
<400> 290
Ala Ser Met Val Gly Leu Asp Ala Gly Val Ala Lys Leu Ala Thr Leu
1 5 10 15
Ser Asp
<210> 291
<211> 23
<212> PRT
<213> Escherichia coli
<400> 291
Leu Ala Arg Leu Gln Arg Gln Leu Ser Arg Lys Val Lys Phe Ser Asn
1 5 10 15
Asn Trp Gln Lys Gln Lys Arg
20
<210> 292
<211> 13
<212> PRT
<213> Escherichia coli
<400> 292
His Ala Met Ile Val Ile Glu Asp Leu Lys Val Ser Asn
1 5 10
<210> 293
<211> 16
<212> PRT
<213> Escherichia coli
<400> 293
Tyr Thr Ala Asn Ala Asp Val Asn Gly Ala Arg Asn Ile Leu Ala Ala
1 5 10 15
<210> 294
<211> 18
<212> PRT
<213> Clostridium botulinum
<400> 294
Asn Lys Lys Val Gly Ile Asp Val Gly Leu Lys Glu Phe Ala Thr Thr
1 5 10 15
Ser Asp
<210> 295
<211> 23
<212> PRT
<213> Clostridium botulinum
<400> 295
Leu Ala Lys Leu Gln Lys Asp Leu Ser Arg Lys Lys Lys Asn Ser Asn
1 5 10 15
Asn Arg Lys Lys Ala Arg Leu
20
<210> 296
<211> 13
<212> PRT
<213> Clostridium botulinum
<400> 296
Asn Gln Ala Ile Val Ile Glu Asn Leu Lys Val Ser Asn
1 5 10
<210> 297
<211> 16
<212> PRT
<213> Clostridium botulinum
<400> 297
Met Ile Met Asp Arg Asp Leu Asn Ala Ser Lys Asn Leu Leu Asn Leu
1 5 10 15
<210> 298
<211> 18
<212> PRT
<213> Acidaminococcus sp.
<400> 298
Met Tyr Tyr Leu Gly Leu Asp Ile Gly Thr Asn Ser Val Gly Tyr Ala
1 5 10 15
Val Thr
<210> 299
<211> 22
<212> PRT
<213> Acidaminococcus sp.
<400> 299
Ala Glu Arg Arg Ser Phe Arg Thr Ser Arg Arg Arg Leu Asp Arg Arg
1 5 10 15
Gln Gln Arg Val Lys Leu
20
<210> 300
<211> 13
<212> PRT
<213> Acidaminococcus sp.
<400> 300
Pro Lys Arg Ile Phe Ile Glu Met Ala Arg Asp Gly Glu
1 5 10
<210> 301
<211> 16
<212> PRT
<213> Acidaminococcus sp.
<400> 301
Leu His His Ala Lys Asp Ala Phe Leu Ala Ile Val Thr Gly Asn Val
1 5 10 15
<210> 302
<211> 18
<212> PRT
<213> Coprococcus catus
<400> 302
Glu Tyr Phe Leu Gly Leu Asp Met Gly Thr Gly Ser Leu Gly Trp Ala
1 5 10 15
Val Thr
<210> 303
<211> 22
<212> PRT
<213> Coprococcus catus
<400> 303
Glu Glu Arg Arg Met Phe Arg Thr Ala Arg Arg Arg Leu Asp Arg Arg
1 5 10 15
Asn Trp Arg Ile Gln Val
20
<210> 304
<211> 13
<212> PRT
<213> Coprococcus catus
<400> 304
Pro Lys Arg Val Phe Val Glu Met Ala Arg Glu Lys Gln
1 5 10
<210> 305
<211> 16
<212> PRT
<213> Coprococcus catus
<400> 305
Leu His His Ala Lys Asp Ala Tyr Leu Asn Ile Val Val Gly Asn Ala
1 5 10 15
<210> 306
<211> 18
<212> PRT
<213> Treponema denticola
<400> 306
Asp Tyr Phe Leu Gly Leu Asp Val Gly Thr Gly Ser Val Gly Trp Ala
1 5 10 15
Val Thr
<210> 307
<211> 22
<212> PRT
<213> Treponema denticola
<400> 307
Glu Val Arg Arg Leu His Arg Gly Ala Arg Arg Arg Ile Glu Arg Arg
1 5 10 15
Lys Lys Arg Ile Lys Leu
20
<210> 308
<211> 13
<212> PRT
<213> Treponema denticola
<400> 308
Pro Lys Lys Ile Phe Ile Glu Met Ala Lys Gly Ala Glu
1 5 10
<210> 309
<211> 16
<212> PRT
<213> Treponema denticola
<400> 309
Phe His His Ala His Asp Ala Tyr Leu Asn Ile Val Val Gly Asn Val
1 5 10 15
<210> 310
<211> 18
<212> PRT
<213> Mycoplasma mobile
<400> 310
Lys Val Val Leu Gly Leu Asp Leu Gly Ile Ala Ser Val Gly Trp Cys
1 5 10 15
Leu Thr
<210> 311
<211> 22
<212> PRT
<213> Mycoplasma mobile
<400> 311
Glu Thr Arg Arg Lys Lys Arg Gly Gln Arg Arg Arg Asn Arg Arg Leu
1 5 10 15
Phe Thr Arg Lys Arg Asp
20
<210> 312
<211> 13
<212> PRT
<213> Mycoplasma mobile
<400> 312
Ile Glu Lys Ile Val Val Glu Val Thr Arg Ser Ser Asn
1 5 10
<210> 313
<211> 16
<212> PRT
<213> Mycoplasma mobile
<400> 313
Gly His His Ala Glu Asp Ala Tyr Phe Ile Thr Ile Ile Ser Gln Tyr
1 5 10 15
<210> 314
<211> 18
<212> PRT
<213> Streptococcus thermophilus
<400> 314
Asp Leu Val Leu Gly Leu Asp Ile Gly Ile Gly Ser Val Gly Val Gly
1 5 10 15
Ile Leu
<210> 315
<211> 22
<212> PRT
<213> Streptococcus thermophilus
<400> 315
Leu Val Arg Arg Thr Asn Arg Gln Gly Arg Arg Leu Thr Arg Arg Lys
1 5 10 15
Lys His Arg Ile Val Arg
20
<210> 316
<211> 13
<212> PRT
<213> Streptococcus thermophilus
<400> 316
Phe Asp Asn Ile Val Ile Glu Met Ala Arg Glu Thr Asn
1 5 10
<210> 317
<211> 16
<212> PRT
<213> Streptococcus thermophilus
<400> 317
His His His Ala Val Asp Ala Leu Ile Ile Ala Ala Ser Ser Gln Leu
1 5 10 15
<210> 318
<211> 18
<212> PRT
<213> Campylobacter jejuni
<400> 318
Ala Arg Ile Leu Ala Phe Asp Ile Gly Ile Ser Ser Ile Gly Trp Ala
1 5 10 15
Phe Ser
<210> 319
<211> 22
<212> PRT
<213> Campylobacter jejuni
<400> 319
Leu Pro Arg Arg Leu Ala Arg Ser Ala Arg Lys Arg Leu Ala Arg Arg
1 5 10 15
Lys Ala Arg Leu Asn His
20
<210> 320
<211> 13
<212> PRT
<213> Campylobacter jejuni
<400> 320
Val His Lys Ile Asn Ile Glu Leu Ala Arg Glu Val Gly
1 5 10
<210> 321
<211> 16
<212> PRT
<213> Campylobacter jejuni
<400> 321
Leu His His Ala Ile Asp Ala Val Ile Ile Ala Tyr Ala Asn Asn Ser
1 5 10 15
<210> 322
<211> 18
<212> PRT
<213> Clostridium perfringens
<400> 322
Asn Tyr Ala Leu Gly Leu Asp Ile Gly Ile Thr Ser Val Gly Trp Ala
1 5 10 15
Val Ile
<210> 323
<211> 22
<212> PRT
<213> Clostridium perfringens
<400> 323
Leu Pro Arg Arg Leu Ala Arg Gly Arg Arg Arg Leu Leu Arg Arg Lys
1 5 10 15
Ala Tyr Arg Val Glu Arg
20
<210> 324
<211> 13
<212> PRT
<213> Clostridium perfringens
<400> 324
Pro Val Arg Ile Asn Ile Glu Leu Ala Arg Asp Leu Ala
1 5 10
<210> 325
<211> 16
<212> PRT
<213> Clostridium perfringens
<400> 325
Lys His His Ala Leu Asp Ala Ala Val Val Gly Val Thr Thr Gln Gly
1 5 10 15
<210> 326
<211> 18
<212> PRT
<213> Akkermansia muciniphila
<400> 326
Ser Leu Thr Phe Ser Phe Asp Ile Gly Tyr Ala Ser Ile Gly Trp Ala
1 5 10 15
Val Ile
<210> 327
<211> 22
<212> PRT
<213> Akkermansia muciniphila
<400> 327
Phe Lys Arg Arg Glu Tyr Arg Arg Leu Arg Arg Asn Ile Arg Ser Arg
1 5 10 15
Arg Val Arg Ile Glu Arg
20
<210> 328
<211> 13
<212> PRT
<213> Akkermansia muciniphila
<400> 328
Ile Ser Arg Val Cys Val Glu Val Gly Lys Glu Leu Thr
1 5 10
<210> 329
<211> 16
<212> PRT
<213> Akkermansia muciniphila
<400> 329
Leu His His Ala Leu Asp Ala Cys Val Leu Gly Leu Ile Pro Tyr Ile
1 5 10 15
<210> 330
<211> 18
<212> PRT
<213> Bifidobacterium longum
<400> 330
Arg Tyr Arg Ile Gly Ile Asp Val Gly Leu Asn Ser Val Gly Leu Ala
1 5 10 15
Ala Val
<210> 331
<211> 22
<212> PRT
<213> Bifidobacterium longum
<400> 331
Asn Met Ser Gly Val Ala Arg Arg Thr Arg Arg Met Arg Arg Arg Lys
1 5 10 15
Arg Glu Arg Leu His Lys
20
<210> 332
<211> 13
<212> PRT
<213> Bifidobacterium longum
<400> 332
Pro Val Ser Val Asn Ile Glu His Val Arg Ser Ser Phe
1 5 10
<210> 333
<211> 16
<212> PRT
<213> Bifidobacterium longum
<400> 333
Arg His His Ala Val Asp Ala Ser Val Ile Ala Met Met Asn Thr Ala
1 5 10 15
<210> 334
<211> 18
<212> PRT
<213> Wolinella succinogenes
<400> 334
Val Ser Pro Ile Ser Val Asp Leu Gly Gly Lys Asn Thr Gly Phe Phe
1 5 10 15
Ser Phe
<210> 335
<211> 22
<212> PRT
<213> Wolinella succinogenes
<400> 335
Val Gly Arg Arg Ser Lys Arg His Ser Lys Arg Asn Asn Leu Arg Asn
1 5 10 15
Lys Leu Val Lys Arg Leu
20
<210> 336
<211> 13
<212> PRT
<213> Wolinella succinogenes
<400> 336
Lys Val Pro Ile Ile Leu Glu Gln Asn Ala Phe Glu Tyr
1 5 10
<210> 337
<211> 16
<212> PRT
<213> Wolinella succinogenes
<400> 337
Ser Ser His Ala Ile Asp Ala Val Met Ala Phe Val Ala Arg Tyr Gln
1 5 10 15
<210> 338
<211> 18
<212> PRT
<213> Legionella pneumophila
<400> 338
Leu Ser Pro Ile Gly Ile Asp Leu Gly Gly Lys Phe Thr Gly Val Cys
1 5 10 15
Leu Ser
<210> 339
<211> 22
<212> PRT
<213> Legionella pneumophila
<400> 339
Ala Gln Arg Arg Ala Thr Arg His Arg Val Arg Asn Lys Lys Arg Asn
1 5 10 15
Gln Phe Val Lys Arg Val
20
<210> 340
<211> 13
<212> PRT
<213> Legionella pneumophila
<400> 340
Leu Ile Pro Ile Tyr Leu Glu Gln Asn Arg Phe Glu Phe
1 5 10
<210> 341
<211> 15
<212> PRT
<213> Legionella pneumophila
<400> 341
Pro Ser His Ala Ile Asp Ala Thr Leu Thr Met Ser Ile Gly Leu
1 5 10 15
<210> 342
<211> 18
<212> PRT
<213> Francisella tularensis
<400> 342
Ile Leu Pro Ile Ala Ile Asp Leu Gly Val Lys Asn Thr Gly Val Phe
1 5 10 15
Ser Ala
<210> 343
<211> 22
<212> PRT
<213> Francisella tularensis
<400> 343
Asn Asn Arg Thr Ala Arg Arg His Gln Arg Arg Gly Ile Asp Arg Lys
1 5 10 15
Gln Leu Val Lys Arg Leu
20
<210> 344
<211> 13
<212> PRT
<213> Francisella tularensis
<400> 344
His Ile Pro Ile Ile Thr Glu Ser Asn Ala Phe Glu Phe
1 5 10
<210> 345
<211> 16
<212> PRT
<213> Francisella tularensis
<400> 345
Tyr Ser His Leu Ile Asp Ala Met Leu Ala Phe Cys Ile Ala Ala Asp
1 5 10 15
<210> 346
<211> 18
<212> PRT
<213> Streptococcus pyogenes
<400> 346
Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val Gly Trp Ala
1 5 10 15
Val Ile
<210> 347
<211> 22
<212> PRT
<213> Streptococcus pyogenes
<400> 347
Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg
1 5 10 15
Lys Asn Arg Ile Cys Tyr
20
<210> 348
<211> 13
<212> PRT
<213> Streptococcus pyogenes
<400> 348
Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
1 5 10
<210> 349
<211> 16
<212> PRT
<213> Streptococcus pyogenes
<400> 349
Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val Val Gly Thr Ala
1 5 10 15
<210> 350
<211> 18
<212> PRT
<213> Lactobacillus delbrueckii
<400> 350
Lys Val Ser Leu Gly Val Asp Thr Gly Gln Arg His Ile Gly Phe Ala
1 5 10 15
Ile Val
<210> 351
<211> 23
<212> PRT
<213> Lactobacillus delbrueckii
<400> 351
Tyr Thr Arg Lys Ile Tyr Arg Arg Ser Lys Arg Asn Arg Lys Thr Arg
1 5 10 15
Tyr Arg Gln Ala Arg Phe Leu
20
<210> 352
<211> 13
<212> PRT
<213> Lactobacillus delbrueckii
<400> 352
Asn Pro Asp Leu His Ile Glu Val Gly Lys Phe Asp Met
1 5 10
<210> 353
<211> 15
<212> PRT
<213> Lactobacillus delbrueckii
<400> 353
Lys Gly His Phe Met Asp Ala Ile Ala Ile Ser Gly Ile Lys Pro
1 5 10 15
<210> 354
<211> 18
<212> PRT
<213> Methanohalobium evestigatum
<400> 354
Pro Val Val Ala Gly Met Asp Ser Gly Ser Lys His Ile Gly Cys Ala
1 5 10 15
Ala Val
<210> 355
<211> 23
<212> PRT
<213> Methanohalobium evestigatum
<400> 355
Lys Asp Arg Ala Asp Tyr Arg Arg Asn Arg Arg Ser Arg Lys Thr Arg
1 5 10 15
Tyr Arg Lys Pro Arg Phe Asp
20
<210> 356
<211> 13
<212> PRT
<213> Methanohalobium evestigatum
<400> 356
Val Lys Lys Trp Ile Val Glu Thr Ala Ser Phe Asp Ile
1 5 10
<210> 357
<211> 15
<212> PRT
<213> Methanohalobium evestigatum
<400> 357
Lys Thr His Tyr Asn Asp Ala Val Ala Ile Cys Cys Asp Glu Asn
1 5 10 15
<210> 358
<211> 18
<212> PRT
<213> Clostridium botulinum
<400> 358
Pro Ile Thr Leu Gly Ile Asp Ser Gly Tyr Leu Asn Ile Gly Phe Ser
1 5 10 15
Ala Ile
<210> 359
<211> 22
<212> PRT
<213> Clostridium botulinum
<400> 359
Lys Glu Lys Ala Met Tyr Arg Arg Gln Arg Arg Ser Arg Leu Arg Tyr
1 5 10 15
Arg Lys Pro Arg Phe Asn
20
<210> 360
<211> 13
<212> PRT
<213> Clostridium botulinum
<400> 360
Ile Thr Asn Ile Ile Ile Glu Val Ala Asn Phe Asp Thr
1 5 10
<210> 361
<211> 15
<212> PRT
<213> Clostridium botulinum
<400> 361
Lys Thr His Tyr Asn Asp Ala Phe Cys Ile Ala Gly Ser Ser Asn
1 5 10 15
<210> 362
<211> 18
<212> PRT
<213> Geobacillus thermoleovorans
<400> 362
Pro Val Ser Leu Gly Val Asp Met Gly Thr Arg His Val Gly Ile Ser
1 5 10 15
Ala Thr
<210> 363
<211> 23
<212> PRT
<213> Geobacillus thermoleovorans
<400> 363
Ala Ile Arg Arg Gln Phe Arg Arg Ser Arg Arg Asn Arg Lys Thr Arg
1 5 10 15
Tyr Arg Glu Ala Arg Phe Leu
20
<210> 364
<211> 13
<212> PRT
<213> Geobacillus thermoleovorans
<400> 364
Val Thr Ser Val Thr Ile Glu Val Ala Ala Phe Asp Thr
1 5 10
<210> 365
<211> 15
<212> PRT
<213> Geobacillus thermoleovorans
<400> 365
Lys Ser His Met Val Asp Ala Arg Cys Ile Ser Gly Asn Pro Leu
1 5 10 15
<210> 366
<211> 18
<212> PRT
<213> Ammonifex degensii
<400> 366
Ser Leu Arg Ala Lys Val Asp Asp Gly Ser Arg Tyr Val Gly Ile Ala
1 5 10 15
Leu Val
<210> 367
<211> 23
<212> PRT
<213> Ammonifex degensii
<400> 367
Thr Leu Arg Arg Glu Tyr Arg Arg Gly Arg Arg Tyr Arg Ile Val Arg
1 5 10 15
His Arg Pro Cys Arg Asn Arg
20
<210> 368
<211> 13
<212> PRT
<213> Ammonifex degensii
<400> 368
Ile Ser Gly Val Asp Val Glu Leu Val Ser Ser Gly Val
1 5 10
<210> 369
<211> 15
<212> PRT
<213> Ammonifex degensii
<400> 369
Lys Ser His Thr Asn Asp Ala Leu Ser Leu Phe Leu Pro Gly Gly
1 5 10 15
<210> 370
<211> 18
<212> PRT
<213> Polaromonas sp.
<400> 370
Pro Leu Arg Ile Lys Leu Asp Pro Gly Ser Lys Thr Thr Gly Val Ala
1 5 10 15
Leu Val
<210> 371
<211> 22
<212> PRT
<213> Polaromonas sp.
<400> 371
Thr Ala Arg Arg Gln Met Arg Arg Arg Arg Arg Ser Asn Leu Arg Cys
1 5 10 15
Arg Ala Pro Arg Phe Leu
20
<210> 372
<211> 13
<212> PRT
<213> Polaromonas sp.
<400> 372
Val Arg Ala Ile Ser Ser Glu Leu Val Arg Phe Asp Met
1 5 10
<210> 373
<211> 15
<212> PRT
<213> Polaromonas sp.
<400> 373
Lys Thr His Ala Leu Asp Ala Ala Cys Val Gly Gln Val Arg Phe
1 5 10 15
<210> 374
<211> 18
<212> PRT
<213> Anabaena variabilis
<400> 374
Asp Leu Arg Ile Lys Leu Asp Pro Gly Ala Lys Ile Thr Gly Ile Ala
1 5 10 15
Leu Val
<210> 375
<211> 23
<212> PRT
<213> Anabaena variabilis
<400> 375
Ile Ser Arg Arg Gln Leu Arg Arg Thr Arg Arg Asn Arg Lys Thr Arg
1 5 10 15
Tyr Arg Lys Pro Arg Phe Leu
20
<210> 376
<211> 13
<212> PRT
<213> Anabaena variabilis
<400> 376
Ile Thr Ala Ile Ser Thr Glu Leu Val Lys Phe Asp Met
1 5 10
<210> 377
<211> 15
<212> PRT
<213> Anabaena variabilis
<400> 377
Lys Thr His Trp Leu Asp Ala Ala Cys Val Gly Gln Ser Thr Pro
1 5 10 15
<210> 378
<211> 18
<212> PRT
<213> Nostoc sp.
<400> 378
Pro Leu Arg Leu Lys Phe Asp Pro Gly Ala Lys Tyr Thr Gly Ile Ala
1 5 10 15
Leu Val
<210> 379
<211> 23
<212> PRT
<213> Nostoc sp.
<400> 379
Thr Ser Arg Arg Gln Leu Arg Arg Ser Arg Arg Ser Arg Lys Thr Arg
1 5 10 15
Tyr Arg Gln Pro Arg Phe Phe
20
<210> 380
<211> 13
<212> PRT
<213> Nostoc sp.
<400> 380
Ile Thr Ala Ile Ser Gln Glu Leu Val Lys Phe Asp Thr
1 5 10
<210> 381
<211> 15
<212> PRT
<213> Nostoc sp.
<400> 381
Lys Ser His Trp Leu Asp Ala Cys Cys Val Gly Ala Ser Thr Pro
1 5 10 15
<210> 382
<211> 18
<212> PRT
<213> Thermus thermophilus
<400> 382
Met Val Val Ala Gly Ile Asp Pro Gly Ile Thr His Leu Gly Leu Gly
1 5 10 15
Val Val
<210> 383
<211> 13
<212> PRT
<213> Thermus thermophilus
<400> 383
Pro Glu Ala Val Ala Val Glu Glu Gln Phe Phe Tyr Arg
1 5 10
<210> 384
<211> 15
<212> PRT
<213> Thermus thermophilus
<400> 384
Pro Ser His Leu Ala Asp Ala Leu Ala Ile Ala Leu Thr His Ala
1 5 10 15
<210> 385
<211> 18
<212> PRT
<213> Thermus thermophilus
<400> 385
Met Val Val Ala Gly Ile Asp Pro Gly Ile Thr His Leu Gly Leu Gly
1 5 10 15
Val Val
<210> 386
<211> 13
<212> PRT
<213> Thermus thermophilus
<400> 386
Pro Glu Ala Val Ala Val Glu Glu Gln Phe Phe Tyr Arg
1 5 10
<210> 387
<211> 15
<212> PRT
<213> Thermus thermophilus
<400> 387
Pro Ser His Leu Ala Asp Ala Leu Ala Ile Ala Leu Thr His Ala
1 5 10 15
<210> 388
<211> 18
<212> PRT
<213> Campylobacter jejuni
<400> 388
Ala Arg Ile Leu Ala Phe Asp Ile Gly Ile Ser Ser Ile Gly Trp Ala
1 5 10 15
Phe Ser
<210> 389
<211> 22
<212> PRT
<213> Campylobacter jejuni
<400> 389
Leu Pro Arg Arg Leu Ala Arg Ser Ala Arg Lys Arg Leu Ala Arg Arg
1 5 10 15
Lys Ala Arg Leu Asn His
20
<210> 390
<211> 13
<212> PRT
<213> Campylobacter jejuni
<400> 390
Val His Lys Ile Asn Ile Glu Leu Ala Arg Glu Val Gly
1 5 10
<210> 391
<211> 16
<212> PRT
<213> Campylobacter jejuni
<400> 391
Leu His His Ala Ile Asp Ala Val Ile Ile Ala Tyr Ala Asn Asn Ser
1 5 10 15
<210> 392
<211> 18
<212> PRT
<213> Clostridium perfringens
<400> 392
Asn Tyr Ala Leu Gly Leu Asp Ile Gly Ile Thr Ser Val Gly Trp Ala
1 5 10 15
Val Ile
<210> 393
<211> 22
<212> PRT
<213> Clostridium perfringens
<400> 393
Leu Pro Arg Arg Leu Ala Arg Gly Arg Arg Arg Leu Leu Arg Arg Lys
1 5 10 15
Ala Tyr Arg Val Glu Arg
20
<210> 394
<211> 13
<212> PRT
<213> Clostridium perfringens
<400> 394
Pro Val Arg Ile Asn Ile Glu Leu Ala Arg Asp Leu Ala
1 5 10
<210> 395
<211> 16
<212> PRT
<213> Clostridium perfringens
<400> 395
Lys His His Ala Leu Asp Ala Ala Val Val Gly Val Thr Thr Gln Gly
1 5 10 15
<210> 396
<211> 18
<212> PRT
<213> Akkermansia muciniphila
<400> 396
Ser Leu Thr Phe Ser Phe Asp Ile Gly Tyr Ala Ser Ile Gly Trp Ala
1 5 10 15
Val Ile
<210> 397
<211> 22
<212> PRT
<213> Akkermansia muciniphila
<400> 397
Phe Lys Arg Arg Glu Tyr Arg Arg Leu Arg Arg Asn Ile Arg Ser Arg
1 5 10 15
Arg Val Arg Ile Glu Arg
20
<210> 398
<211> 13
<212> PRT
<213> Akkermansia muciniphila
<400> 398
Ile Ser Arg Val Cys Val Glu Val Gly Lys Glu Leu Thr
1 5 10
<210> 399
<211> 16
<212> PRT
<213> Akkermansia muciniphila
<400> 399
Leu His His Ala Leu Asp Ala Cys Val Leu Gly Leu Ile Pro Tyr Ile
1 5 10 15
<210> 400
<211> 18
<212> PRT
<213> Bifidobacterium longum
<400> 400
Arg Tyr Arg Ile Gly Ile Asp Val Gly Leu Asn Ser Val Gly Leu Ala
1 5 10 15
Ala Val
<210> 401
<211> 22
<212> PRT
<213> Bifidobacterium longum
<400> 401
Asn Met Ser Gly Val Ala Arg Arg Thr Arg Arg Met Arg Arg Arg Lys
1 5 10 15
Arg Glu Arg Leu His Lys
20
<210> 402
<211> 13
<212> PRT
<213> Bifidobacterium longum
<400> 402
Pro Val Ser Val Asn Ile Glu His Val Arg Ser Ser Phe
1 5 10
<210> 403
<211> 16
<212> PRT
<213> Bifidobacterium longum
<400> 403
Arg His His Ala Val Asp Ala Ser Val Ile Ala Met Met Asn Thr Ala
1 5 10 15
<210> 404
<211> 18
<212> PRT
<213> Wolinella succinogenes
<400> 404
Val Ser Pro Ile Ser Val Asp Leu Gly Gly Lys Asn Thr Gly Phe Phe
1 5 10 15
Ser Phe
<210> 405
<211> 22
<212> PRT
<213> Wolinella succinogenes
<400> 405
Val Gly Arg Arg Ser Lys Arg His Ser Lys Arg Asn Asn Leu Arg Asn
1 5 10 15
Lys Leu Val Lys Arg Leu
20
<210> 406
<211> 13
<212> PRT
<213> Wolinella succinogenes
<400> 406
Lys Val Pro Ile Ile Leu Glu Gln Asn Ala Phe Glu Tyr
1 5 10
<210> 407
<211> 16
<212> PRT
<213> Wolinella succinogenes
<400> 407
Ser Ser His Ala Ile Asp Ala Val Met Ala Phe Val Ala Arg Tyr Gln
1 5 10 15
<210> 408
<211> 18
<212> PRT
<213> Legionella pneumophila
<400> 408
Leu Ser Pro Ile Gly Ile Asp Leu Gly Gly Lys Phe Thr Gly Val Cys
1 5 10 15
Leu Ser
<210> 409
<211> 22
<212> PRT
<213> Legionella pneumophila
<400> 409
Ala Gln Arg Arg Ala Thr Arg His Arg Val Arg Asn Lys Lys Arg Asn
1 5 10 15
Gln Phe Val Lys Arg Val
20
<210> 410
<211> 13
<212> PRT
<213> Legionella pneumophila
<400> 410
Leu Ile Pro Ile Tyr Leu Glu Gln Asn Arg Phe Glu Phe
1 5 10
<210> 411
<211> 15
<212> PRT
<213> Legionella pneumophila
<400> 411
Pro Ser His Ala Ile Asp Ala Thr Leu Thr Met Ser Ile Gly Leu
1 5 10 15
<210> 412
<211> 18
<212> PRT
<213> Francisella tularensis
<400> 412
Ile Leu Pro Ile Ala Ile Asp Leu Gly Val Lys Asn Thr Gly Val Phe
1 5 10 15
Ser Ala
<210> 413
<211> 22
<212> PRT
<213> Francisella tularensis
<400> 413
Asn Asn Arg Thr Ala Arg Arg His Gln Arg Arg Gly Ile Asp Arg Lys
1 5 10 15
Gln Leu Val Lys Arg Leu
20
<210> 414
<211> 13
<212> PRT
<213> Francisella tularensis
<400> 414
His Ile Pro Ile Ile Thr Glu Ser Asn Ala Phe Glu Phe
1 5 10
<210> 415
<211> 16
<212> PRT
<213> Francisella tularensis
<400> 415
Tyr Ser His Leu Ile Asp Ala Met Leu Ala Phe Cys Ile Ala Ala Asp
1 5 10 15
<210> 416
<211> 18
<212> PRT
<213> Streptococcus pyogenes
<400> 416
Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val Gly Trp Ala
1 5 10 15
Val Ile
<210> 417
<211> 22
<212> PRT
<213> Streptococcus pyogenes
<400> 417
Glu Ala Thr Arg Leu Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg
1 5 10 15
Lys Asn Arg Ile Cys Tyr
20
<210> 418
<211> 13
<212> PRT
<213> Streptococcus pyogenes
<400> 418
Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
1 5 10
<210> 419
<211> 16
<212> PRT
<213> Streptococcus pyogenes
<400> 419
Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val Val Gly Thr Ala
1 5 10 15
<210> 420
<211> 18
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Candidatus Methanomethylophilus
alvus sequence"
<400> 420
Leu Lys Ile Ile Gly Ile Asp Arg Gly Glu Arg Asn Leu Ile Tyr Val
1 5 10 15
Thr Met
<210> 421
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Candidatus Methanomethylophilus
alvus sequence"
<400> 421
Arg Lys Ala Leu Asp Val Arg Glu Tyr Asp Asn Lys Glu Ala Arg Arg
1 5 10 15
Asn Trp Thr Lys Val Glu
20
<210> 422
<211> 13
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(13)
<223> /note="Description of Unknown: Candidatus Methanomethylophilus
alvus sequence"
<400> 422
Asn Ala Ile Ile Val Met Glu Asp Leu Asn His Gly Phe
1 5 10
<210> 423
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Candidatus Methanomethylophilus
alvus sequence"
<400> 423
Leu Pro Gln Asp Ser Asp Ala Asn Gly Ala Tyr Asn Ile Ala Leu Lys
1 5 10 15
<210> 424
<211> 18
<212> PRT
<213> Synergistes jonesii
<400> 424
Val Asn Ile Ile Gly Ile Asp Arg Gly Glu Arg Asn Leu Val Tyr Val
1 5 10 15
Ser Leu
<210> 425
<211> 22
<212> PRT
<213> Synergistes jonesii
<400> 425
His Ala Lys Leu Asn Gln Lys Glu Lys Glu Arg Asp Thr Ala Arg Lys
1 5 10 15
Ser Trp Lys Thr Ile Gly
20
<210> 426
<211> 13
<212> PRT
<213> Synergistes jonesii
<400> 426
Asn Ala Val Ile Val Met Glu Asp Leu Asn Ile Gly Phe
1 5 10
<210> 427
<211> 16
<212> PRT
<213> Synergistes jonesii
<400> 427
Leu Pro Ile Asp Ala Asp Ala Asn Gly Ala Tyr His Ile Ala Leu Lys
1 5 10 15
<210> 428
<211> 18
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 428
Pro Tyr Val Ile Gly Ile Asp Arg Gly Glu Arg Asn Leu Leu Tyr Ile
1 5 10 15
Val Val
<210> 429
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (2)..(22)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 429
His Ser Leu Leu Asp Lys Lys Glu Lys Glu Arg Phe Glu Ala Arg Gln
1 5 10 15
Asn Trp Thr Ser Ile Glu
20
<210> 430
<211> 13
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(13)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 430
Asp Ala Val Ile Ala Leu Glu Asp Leu Asn Ser Gly Phe
1 5 10
<210> 431
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 431
Leu Pro Lys Asn Ala Asp Ala Asn Gly Ala Tyr Asn Ile Ala Arg Lys
1 5 10 15
<210> 432
<211> 18
<212> PRT
<213> Francisella tularensis
<400> 432
Val His Ile Leu Ser Ile Asp Arg Gly Glu Arg His Leu Ala Tyr Tyr
1 5 10 15
Thr Leu
<210> 433
<211> 22
<212> PRT
<213> Francisella tularensis
<400> 433
His Asp Lys Leu Ala Ala Ile Glu Lys Asp Arg Asp Ser Ala Arg Lys
1 5 10 15
Asp Trp Lys Lys Ile Asn
20
<210> 434
<211> 13
<212> PRT
<213> Francisella tularensis
<400> 434
Asn Ala Ile Val Val Phe Glu Asp Leu Asn Phe Gly Phe
1 5 10
<210> 435
<211> 16
<212> PRT
<213> Francisella tularensis
<400> 435
Met Pro Gln Asp Ala Asp Ala Asn Gly Ala Tyr His Ile Gly Leu Lys
1 5 10 15
<210> 436
<211> 18
<212> PRT
<213> Moraxella caprae
<400> 436
Val Asn Val Ile Gly Ile Asp Arg Gly Glu Arg His Leu Leu Tyr Leu
1 5 10 15
Thr Val
<210> 437
<211> 22
<212> PRT
<213> Moraxella caprae
<400> 437
His Lys Ile Leu Asp Lys Arg Glu Ile Glu Arg Leu Asn Ala Arg Val
1 5 10 15
Gly Trp Gly Glu Ile Glu
20
<210> 438
<211> 13
<212> PRT
<213> Moraxella caprae
<400> 438
Asn Ala Ile Val Val Leu Glu Asp Leu Asn Phe Gly Phe
1 5 10
<210> 439
<211> 16
<212> PRT
<213> Moraxella caprae
<400> 439
Gln Pro Gln Asn Ala Asp Ala Asn Gly Ala Tyr His Ile Ala Leu Lys
1 5 10 15
<210> 440
<211> 18
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 440
Met His Ile Ile Gly Ile Asp Arg Gly Glu Arg Asn Leu Ile Tyr Leu
1 5 10 15
Cys Met
<210> 441
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 441
His Gln Leu Leu Lys Thr Arg Glu Asp Glu Asn Lys Ser Ala Arg Gln
1 5 10 15
Ser Trp Gln Thr Ile His
20
<210> 442
<211> 13
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(3)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 442
Asn Ala Ile Val Val Leu Glu Asp Leu Asn Phe Gly Phe
1 5 10
<210> 443
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 443
Met Pro Leu Asp Ala Asp Ala Asn Gly Ala Tyr Asn Ile Ala Arg Lys
1 5 10 15
<210> 444
<211> 18
<212> PRT
<213> Prevotella albensis
<400> 444
Thr His Ile Ile Gly Ile Asp Arg Gly Glu Arg His Leu Leu Tyr Leu
1 5 10 15
Ser Leu
<210> 445
<211> 22
<212> PRT
<213> Prevotella albensis
<400> 445
His Asn Leu Leu Glu Lys Arg Glu Lys Glu Arg Thr Glu Ala Arg His
1 5 10 15
Ser Trp Ser Ser Ile Glu
20
<210> 446
<211> 13
<212> PRT
<213> Prevotella albensis
<400> 446
Asn Ala Ile Val Val Leu Glu Asp Leu Asn Gly Gly Phe
1 5 10
<210> 447
<211> 16
<212> PRT
<213> Prevotella albensis
<400> 447
Phe Pro Glu Asn Ala Asp Ala Asn Gly Ala Tyr Asn Ile Ala Arg Lys
1 5 10 15
<210> 448
<211> 18
<212> PRT
<213> Smithella sp.
<400> 448
Ile Asn Ile Ile Gly Ile Asp Arg Gly Glu Arg His Leu Leu Tyr Tyr
1 5 10 15
Ala Leu
<210> 449
<211> 22
<212> PRT
<213> Smithella sp.
<400> 449
His Asn Leu Leu Asp Lys Lys Glu Gly Asp Arg Ala Thr Ala Arg Gln
1 5 10 15
Glu Trp Gly Val Ile Glu
20
<210> 450
<211> 13
<212> PRT
<213> Smithella sp.
<400> 450
Asn Ala Ile Ile Val Met Glu Asp Leu Asn Phe Gly Phe
1 5 10
<210> 451
<211> 16
<212> PRT
<213> Smithella sp.
<400> 451
Met Pro Lys Asn Ala Asp Ala Asn Gly Ala Tyr His Ile Ala Leu Lys
1 5 10 15
<210> 452
<211> 18
<212> PRT
<213> Porphyromonas crevioricanis
<400> 452
Met His Val Ile Gly Ile Asp Arg Gly Glu Arg Asn Leu Leu Tyr Ile
1 5 10 15
Cys Val
<210> 453
<211> 22
<212> PRT
<213> Porphyromonas crevioricanis
<400> 453
His Asp Leu Leu Glu Ser Arg Asp Lys Asp Arg Gln Gln Glu Arg Arg
1 5 10 15
Asn Trp Gln Thr Ile Glu
20
<210> 454
<211> 13
<212> PRT
<213> Porphyromonas crevioricanis
<400> 454
Lys Ala Val Val Ala Leu Glu Asp Leu Asn Met Gly Phe
1 5 10
<210> 455
<211> 16
<212> PRT
<213> Porphyromonas crevioricanis
<400> 455
Leu Pro Lys Asp Ala Asp Ala Asn Gly Ala Tyr Asn Ile Ala Leu Lys
1 5 10 15
<210> 456
<211> 18
<212> PRT
<213> Alicyclobacillus acidoterrestris
<400> 456
Leu Arg Val Met Ser Val Asp Leu Gly Leu Arg Thr Ser Ala Ser Ile
1 5 10 15
Ser Val
<210> 457
<211> 22
<212> PRT
<213> Alicyclobacillus acidoterrestris
<400> 457
Gln Arg Thr Leu Arg Gln Leu Arg Thr Gln Leu Ala Tyr Leu Arg Leu
1 5 10 15
Leu Val Arg Cys Gly Ser
20
<210> 458
<211> 12
<212> PRT
<213> Alicyclobacillus acidoterrestris
<400> 458
Cys Gln Leu Ile Leu Leu Glu Glu Leu Ser Glu Tyr
1 5 10
<210> 459
<211> 16
<212> PRT
<213> Alicyclobacillus acidoterrestris
<400> 459
His Gln Ile His Ala Asp Leu Asn Ala Ala Gln Asn Leu Gln Gln Arg
1 5 10 15
<210> 460
<211> 18
<212> PRT
<213> Alicyclobacillus contaminans
<400> 460
Val Arg Val Met Ser Val Asp Leu Gly Val Arg Tyr Gly Ala Ala Ile
1 5 10 15
Ser Val
<210> 461
<211> 22
<212> PRT
<213> Alicyclobacillus contaminans
<400> 461
Lys Gln Ala Leu Ala Ala Ile Arg Ala Glu Met Ser Ile Leu Arg Lys
1 5 10 15
Trp Leu Arg Val Ser Gln
20
<210> 462
<211> 12
<212> PRT
<213> Alicyclobacillus contaminans
<400> 462
Cys Asp Leu Ile Leu Phe Glu Asp Leu Ser Arg Tyr
1 5 10
<210> 463
<211> 16
<212> PRT
<213> Alicyclobacillus contaminans
<400> 463
Lys Cys Val His Ala Asp Ile Asn Ala Ala His Asn Leu Gln Arg Arg
1 5 10 15
<210> 464
<211> 18
<212> PRT
<213> Desulfovibrio inopinatus
<400> 464
Leu Arg Val Leu Ser Val Asp Leu Gly Met Arg Thr Phe Ala Ser Cys
1 5 10 15
Ser Val
<210> 465
<211> 22
<212> PRT
<213> Desulfovibrio inopinatus
<400> 465
Arg Ala Glu Ile Tyr Ala Leu Lys Arg Asp Ile Gln Arg Leu Lys Ser
1 5 10 15
Leu Leu Arg Leu Gly Glu
20
<210> 466
<211> 12
<212> PRT
<213> Desulfovibrio inopinatus
<400> 466
Cys Gln Leu Ile Leu Phe Glu Asp Leu Ala Arg Tyr
1 5 10
<210> 467
<211> 16
<212> PRT
<213> Desulfovibrio inopinatus
<400> 467
Cys Val Ile His Ala Asp Met Asn Ala Ala Gln Asn Leu Gln Arg Arg
1 5 10 15
<210> 468
<211> 18
<212> PRT
<213> Desulfonatronum thiodismutans
<400> 468
Leu Arg Val Leu Ser Val Asp Leu Gly Val Arg Ser Phe Ala Ala Cys
1 5 10 15
Ser Val
<210> 469
<211> 22
<212> PRT
<213> Desulfonatronum thiodismutans
<400> 469
Met Glu Glu Leu Arg Ser Leu Asn Gly Asp Ile Arg Arg Leu Lys Ala
1 5 10 15
Ile Leu Arg Leu Ser Val
20
<210> 470
<211> 12
<212> PRT
<213> Desulfonatronum thiodismutans
<400> 470
Cys Arg Leu Ile Leu Phe Glu Asp Leu Ala Arg Tyr
1 5 10
<210> 471
<211> 16
<212> PRT
<213> Desulfonatronum thiodismutans
<400> 471
His Val Ile His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln Arg Arg
1 5 10 15
<210> 472
<211> 18
<212> PRT
<213> Tuberibacillus calidus
<400> 472
Leu Arg Val Met Ser Val Asp Leu Gly Gln Arg Gln Ala Ala Ala Ile
1 5 10 15
Ser Ile
<210> 473
<211> 22
<212> PRT
<213> Tuberibacillus calidus
<400> 473
Asp Gln Ala Ile Arg Asp Leu Ser Arg Lys Leu Lys Phe Leu Lys Asn
1 5 10 15
Val Leu Asn Met Gln Lys
20
<210> 474
<211> 12
<212> PRT
<213> Tuberibacillus calidus
<400> 474
Cys Gln Leu Val Leu Phe Glu Asp Leu Ser Arg Tyr
1 5 10
<210> 475
<211> 16
<212> PRT
<213> Tuberibacillus calidus
<400> 475
Val Ile Thr His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln Lys Arg
1 5 10 15
<210> 476
<211> 18
<212> PRT
<213> Bacillus thermoamylovorans
<400> 476
Leu Arg Val Met Ser Ile Asp Leu Gly Gln Arg Gln Ala Ala Ala Ala
1 5 10 15
Ser Ile
<210> 477
<211> 22
<212> PRT
<213> Bacillus thermoamylovorans
<400> 477
Glu Asp Asn Leu Lys Leu Met Asn Gln Lys Leu Asn Phe Leu Arg Asn
1 5 10 15
Val Leu His Phe Gln Gln
20
<210> 478
<211> 12
<212> PRT
<213> Bacillus thermoamylovorans
<400> 478
Cys Gln Ile Ile Leu Phe Glu Asp Leu Ser Asn Tyr
1 5 10
<210> 479
<211> 16
<212> PRT
<213> Bacillus thermoamylovorans
<400> 479
Val Thr Thr His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln Lys Arg
1 5 10 15
<210> 480
<211> 18
<212> PRT
<213> Bacillus sp.
<400> 480
Phe Arg Val Met Ser Ile Asp Leu Gly Leu Arg Ala Ala Ala Ala Thr
1 5 10 15
Ser Ile
<210> 481
<211> 22
<212> PRT
<213> Bacillus sp.
<400> 481
Phe Gln Leu His Gln Arg Val Lys Phe Gln Ile Arg Val Leu Ala Gln
1 5 10 15
Ile Met Arg Met Ala Asn
20
<210> 482
<211> 12
<212> PRT
<213> Bacillus sp.
<400> 482
Cys Gln Val Ile Leu Phe Glu Asn Leu Ser Gln Tyr
1 5 10
<210> 483
<211> 16
<212> PRT
<213> Bacillus sp.
<400> 483
Val Phe Leu Gln Ala Asp Ile Asn Ala Ala His Asn Leu Gln Lys Arg
1 5 10 15
<210> 484
<211> 18
<212> PRT
<213> Methylobacterium nodulans
<400> 484
Leu Arg Val Leu Ser Ile Asp Leu Gly Val Arg Ser Phe Ala Thr Cys
1 5 10 15
Ser Val
<210> 485
<211> 22
<212> PRT
<213> Methylobacterium nodulans
<400> 485
Asp Ala Glu Leu Arg Gln Leu Arg Gly Gly Leu Asn Arg His Arg Gln
1 5 10 15
Leu Leu Arg Ala Ala Thr
20
<210> 486
<211> 12
<212> PRT
<213> Methylobacterium nodulans
<400> 486
Cys His Val Ile Leu Phe Glu Asp Leu Ser Arg Tyr
1 5 10
<210> 487
<211> 16
<212> PRT
<213> Methylobacterium nodulans
<400> 487
Ser Arg Ile His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln Arg Arg
1 5 10 15
<210> 488
<211> 18
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 488
Lys Asn Ile Val Ser Ile Asp Gln Gly Glu Ala Gly Phe Ala Tyr Ala
1 5 10 15
Val Phe
<210> 489
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 489
His Ser Val Lys Lys Tyr Arg Gly Lys Lys Gln Arg Ile Gln Asn Phe
1 5 10 15
Asn Gln Lys Phe Asp Ser
20
<210> 490
<211> 13
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(13)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 490
Asn Ala Phe Pro Ile Leu Glu Lys Gln Val Gly Asn Leu
1 5 10
<210> 491
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 491
Lys Glu Gln His Ala Asp Val Asn Ala Ala Ile Asn Ile Gly Arg Arg
1 5 10 15
<210> 492
<211> 18
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 492
Asp His Ile Val Ala Ile Asp Leu Gly Glu Arg Ser Val Gly Phe Ala
1 5 10 15
Val Phe
<210> 493
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 493
Lys Ala Val Arg Ser His Arg Arg Arg Arg Gln Pro Asn Gln Lys Val
1 5 10 15
Asn Gln Thr Tyr Ser Thr
20
<210> 494
<211> 13
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(13)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 494
Asn Ala Phe Pro Val Leu Glu Phe Gln Ile Lys Asn Phe
1 5 10
<210> 495
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 495
Trp Thr Gly His Ala Asp Glu Asn Ala Ala Ile Asn Ile Gly Arg Arg
1 5 10 15
<210> 496
<211> 18
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Metagenome sequence"
<400> 496
Asp Arg Ile Val Ala Ile Asp Leu Gly Glu Arg Lys Ile Gly Tyr Ala
1 5 10 15
Ile Phe
<210> 497
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Metagenome sequence"
<400> 497
Lys Ala Val Gln Thr His Arg Asn Arg Arg Gln Pro Asn Tyr Arg Ile
1 5 10 15
Asp Gln Thr Tyr Ser Lys
20
<210> 498
<211> 13
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(13)
<223> /note="Description of Unknown: Metagenome sequence"
<400> 498
Gly Gly Phe Pro Val Leu Glu Ser Ser Val Arg Asn Phe
1 5 10
<210> 499
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Metagenome sequence"
<400> 499
His Glu Cys His Ala Asp Glu Asn Ala Ala Ile Asn Ile Gly Arg Lys
1 5 10 15
<210> 500
<211> 18
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 500
Asp His Leu Leu Ala Ile Asp Leu Gly Glu Lys Arg Val Gly Tyr Ala
1 5 10 15
Val Tyr
<210> 501
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 501
Lys Ala Val Arg Ser His Arg Gln Gln Arg Gln Pro Asn Gln Lys Val
1 5 10 15
Asn Gln Thr Tyr Ser Thr
20
<210> 502
<211> 13
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(13)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 502
Asn Ala Phe Pro Val Leu Glu Ser Ser Val Met Asn Phe
1 5 10
<210> 503
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 503
Phe Thr Gly His Ala Asp Glu Asn Ala Ala Ile Asn Ile Gly Trp Lys
1 5 10 15
<210> 504
<211> 15
<212> PRT
<213> Homo sapiens
<400> 504
Ile Pro Asp Trp Ile Val Asp Leu Arg His Glu Leu Thr His Lys
1 5 10 15
<210> 505
<211> 15
<212> PRT
<213> Homo sapiens
<400> 505
Ile Pro Asp Trp Ile Val Asp Leu Arg His Glu Leu Thr His Lys
1 5 10 15
<210> 506
<211> 15
<212> PRT
<213> Mycobacterium tuberculosis
<400> 506
Leu Gly Arg Phe Glu Ser Arg Val Arg Asn Thr Ala Ala His Glu
1 5 10 15
<210> 507
<211> 15
<212> PRT
<213> Mycobacterium tuberculosis
<400> 507
Leu Gly Arg Phe Glu Ser Arg Val Arg Asn Thr Ala Ala His Glu
1 5 10 15
<210> 508
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 508
Trp Ile Arg Ala Gly Trp Arg Ile Arg Asn Arg Ser Ala His Tyr
1 5 10 15
<210> 509
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 509
Trp Ile Arg Ala Gly Trp Phe Ile Arg Asn Arg Ser Ala His Tyr
1 5 10 15
<210> 510
<211> 15
<212> PRT
<213> Thermus thermophilus
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<400> 510
Leu Ala Leu Gly Xaa Val Asp Asp Arg Ser Leu Thr Val His Thr
1 5 10 15
<210> 511
<211> 15
<212> PRT
<213> Thermus thermophilus
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<400> 511
Leu Ala Leu Gly Xaa Val Asp Asp Arg Ser Leu Thr Val His Thr
1 5 10 15
<210> 512
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 512
Leu Lys Ser Met Leu Tyr Ser Met Arg Asn Ser Ser Phe His Phe Ser
1 5 10 15
Thr Glu Asn Val Asp Asn
20
<210> 513
<211> 14
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 513
Phe Arg Asn Glu Ile Asp His Phe His Tyr Phe Tyr Asp Arg
1 5 10
<210> 514
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 514
Leu Lys Asp Val Ile Tyr Ser Met Arg Asn Asp Ser Phe His Tyr Ala
1 5 10 15
Thr Glu Asn His Asn Asn
20
<210> 515
<211> 14
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 515
Leu Arg Asn Tyr Ile Glu His Phe Arg Tyr Tyr Ser Ser Phe
1 5 10
<210> 516
<211> 22
<212> PRT
<213> Clostridium aminophilum
<400> 516
Leu Arg Lys Ala Ile Tyr Ser Leu Arg Asn Glu Thr Phe His Phe Thr
1 5 10 15
Thr Leu Asn Lys Gly Ser
20
<210> 517
<211> 14
<212> PRT
<213> Clostridium aminophilum
<400> 517
Val Arg Lys Tyr Val Asp His Phe Lys Tyr Tyr Ala Thr Ser
1 5 10
<210> 518
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 518
Ile Ile Gln Ile Ile Tyr Ser Leu Arg Asn Lys Ser Phe His Phe Lys
1 5 10 15
Thr Tyr Asp His Gly Asp
20
<210> 519
<211> 14
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 519
Leu Arg Lys Tyr Val Asp His Phe Lys Tyr Tyr Ala Tyr Gly
1 5 10
<210> 520
<211> 22
<212> PRT
<213> Carnobacterium gallinarum
<400> 520
Leu Arg Gly Ser Val Gln Gln Ile Arg Asn Glu Ile Phe His Ser Phe
1 5 10 15
Asp Lys Asn Gln Lys Phe
20
<210> 521
<211> 14
<212> PRT
<213> Carnobacterium gallinarum
<400> 521
Ile Arg Asn Gln Thr Ala His Leu Ser Val Leu Gln Leu Glu
1 5 10
<210> 522
<211> 21
<212> PRT
<213> Carnobacterium gallinarum
<400> 522
Ile Arg Gly Ala Val Gln Arg Val Arg Asn Gln Ile Phe His Gln Gln
1 5 10 15
Ile Asn Lys Arg His
20
<210> 523
<211> 14
<212> PRT
<213> Carnobacterium gallinarum
<400> 523
Ile Arg Asn Asn Ile Ala His Leu His Val Leu Arg Asn Asp
1 5 10
<210> 524
<211> 20
<212> PRT
<213> Paludibacter propionicigenes
<400> 524
Ile Arg Gly Ala Val Gln Gln Ile Arg Asn Asn Val Asn His Tyr Lys
1 5 10 15
Lys Asp Ala Leu
20
<210> 525
<211> 14
<212> PRT
<213> Paludibacter propionicigenes
<400> 525
Ile Arg Asn His Ile Ala His Phe Asn Tyr Leu Thr Lys Asp
1 5 10
<210> 526
<211> 20
<212> PRT
<213> Listeria seeligeri
<400> 526
Leu Arg Gly Ala Ile Ala Pro Ile Arg Asn Glu Ile Ile His Leu Lys
1 5 10 15
Lys His Ser Trp
20
<210> 527
<211> 14
<212> PRT
<213> Listeria seeligeri
<400> 527
Lys Arg Asn Asn Ile Ser His Phe Asn Tyr Leu Asn Gly Gln
1 5 10
<210> 528
<211> 20
<212> PRT
<213> Listeria weihenstephanensis
<400> 528
Ile Arg Gly Ser Ile Gln Gln Ile Arg Asn Glu Val Tyr His Cys Lys
1 5 10 15
Lys His Ser Trp
20
<210> 529
<211> 14
<212> PRT
<213> Listeria weihenstephanensis
<400> 529
Ala Arg Asn His Ile Ala His Leu Asn Tyr Leu Ser Leu Lys
1 5 10
<210> 530
<211> 20
<212> PRT
<213> Listeria newyorkens
<400> 530
Ile Arg Gly Ser Ile Gln Gln Ile Arg Asn Glu Val Tyr His Cys Lys
1 5 10 15
Lys His Ser Trp
20
<210> 531
<211> 14
<212> PRT
<213> Listeria newyorkens
<400> 531
Ala Arg Asn His Ile Ala His Leu Asn Tyr Leu Ser Leu Lys
1 5 10
<210> 532
<211> 21
<212> PRT
<213> Leptotrichia wadei
<400> 532
Ile Ser Tyr Ser Ile Tyr Asn Val Arg Asn Gly Val Gly His Phe Asn
1 5 10 15
Lys Leu Ile Leu Gly
20
<210> 533
<211> 14
<212> PRT
<213> Leptotrichia wadei
<400> 533
Phe Arg Asn Tyr Ile Ala His Phe Leu His Leu His Thr Lys
1 5 10
<210> 534
<211> 21
<212> PRT
<213> Leptotrichia wadei
<400> 534
Met Leu Asn Ala Ile Thr Ser Ile Arg His Arg Val Val His Tyr Asn
1 5 10 15
Met Asn Thr Asn Ser
20
<210> 535
<211> 14
<212> PRT
<213> Leptotrichia wadei
<400> 535
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro Asp Ala
1 5 10
<210> 536
<211> 21
<212> PRT
<213> Leptotrichia wadei
<400> 536
Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe Asn
1 5 10 15
Leu Glu Leu Glu Gly
20
<210> 537
<211> 14
<212> PRT
<213> Leptotrichia wadei
<400> 537
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro His Ala
1 5 10
<210> 538
<211> 22
<212> PRT
<213> Rhodobacter capsulatus
<400> 538
Leu Leu Arg Tyr Leu Arg Gly Cys Arg Asn Gln Thr Phe His Leu Gly
1 5 10 15
Ala Arg Ala Gly Phe Leu
20
<210> 539
<211> 21
<212> PRT
<213> Leptotrichia buccalis
<400> 539
Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe Asn
1 5 10 15
Leu Glu Leu Glu Gly
20
<210> 540
<211> 14
<212> PRT
<213> Rhodobacter capsulatus
<400> 540
Thr Arg Lys Asp Leu Ala His Phe Asn Val Leu Asp Arg Ala
1 5 10
<210> 541
<211> 21
<212> PRT
<213> Leptotrichia sp.
<400> 541
Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe Asn
1 5 10 15
Leu Glu Leu Glu Gly
20
<210> 542
<211> 14
<212> PRT
<213> Leptotrichia buccalis
<400> 542
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro His Ala
1 5 10
<210> 543
<211> 16
<212> PRT
<213> Leptotrichia sp.
<400> 543
Phe Gln Lys Glu Gly Tyr Leu Leu Arg Asn Lys Ile Leu His Asn Ser
1 5 10 15
<210> 544
<211> 14
<212> PRT
<213> Leptotrichia sp.
<400> 544
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro Asn Ala
1 5 10
<210> 545
<211> 15
<212> PRT
<213> Leptotrichia shahii
<400> 545
Phe Thr Lys Ile Gly Thr Asn Glu Arg Asn Arg Ile Leu His Ala
1 5 10 15
<210> 546
<211> 14
<212> PRT
<213> Leptotrichia sp.
<400> 546
Ile Arg Asn Tyr Ile Ser His Phe Tyr Ile Val Arg Asn Pro
1 5 10
<210> 547
<211> 14
<212> PRT
<213> Leptotrichia shahii
<400> 547
Ile Arg Asn Tyr Ile Ser His Phe Tyr Ile Val Arg Asn Pro
1 5 10
<210> 548
<211> 1129
<212> PRT
<213> Alicyclobacillus acidoterrestris
<400> 548
Met Ala Val Lys Ser Ile Lys Val Lys Leu Arg Leu Asp Asp Met Pro
1 5 10 15
Glu Ile Arg Ala Gly Leu Trp Lys Leu His Lys Glu Val Asn Ala Gly
20 25 30
Val Arg Tyr Tyr Thr Glu Trp Leu Ser Leu Leu Arg Gln Glu Asn Leu
35 40 45
Tyr Arg Arg Ser Pro Asn Gly Asp Gly Glu Gln Glu Cys Asp Lys Thr
50 55 60
Ala Glu Glu Cys Lys Ala Glu Leu Leu Glu Arg Leu Arg Ala Arg Gln
65 70 75 80
Val Glu Asn Gly His Arg Gly Pro Ala Gly Ser Asp Asp Glu Leu Leu
85 90 95
Gln Leu Ala Arg Gln Leu Tyr Glu Leu Leu Val Pro Gln Ala Ile Gly
100 105 110
Ala Lys Gly Asp Ala Gln Gln Ile Ala Arg Lys Phe Leu Ser Pro Leu
115 120 125
Ala Asp Lys Asp Ala Val Gly Gly Leu Gly Ile Ala Lys Ala Gly Asn
130 135 140
Lys Pro Arg Trp Val Arg Met Arg Glu Ala Gly Glu Pro Gly Trp Glu
145 150 155 160
Glu Glu Lys Glu Lys Ala Glu Thr Arg Lys Ser Ala Asp Arg Thr Ala
165 170 175
Asp Val Leu Arg Ala Leu Ala Asp Phe Gly Leu Lys Pro Leu Met Arg
180 185 190
Val Tyr Thr Asp Ser Glu Met Ser Ser Val Glu Trp Lys Pro Leu Arg
195 200 205
Lys Gly Gln Ala Val Arg Thr Trp Asp Arg Asp Met Phe Gln Gln Ala
210 215 220
Ile Glu Arg Met Met Ser Trp Glu Ser Trp Asn Gln Arg Val Gly Gln
225 230 235 240
Glu Tyr Ala Lys Leu Val Glu Gln Lys Asn Arg Phe Glu Gln Lys Asn
245 250 255
Phe Val Gly Gln Glu His Leu Val His Leu Val Asn Gln Leu Gln Gln
260 265 270
Asp Met Lys Glu Ala Ser Pro Gly Leu Glu Ser Lys Glu Gln Thr Ala
275 280 285
His Tyr Val Thr Gly Arg Ala Leu Arg Gly Ser Asp Lys Val Phe Glu
290 295 300
Lys Trp Gly Lys Leu Ala Pro Asp Ala Pro Phe Asp Leu Tyr Asp Ala
305 310 315 320
Glu Ile Lys Asn Val Gln Arg Arg Asn Thr Arg Arg Phe Gly Ser His
325 330 335
Asp Leu Phe Ala Lys Leu Ala Glu Pro Glu Tyr Gln Ala Leu Trp Arg
340 345 350
Glu Asp Ala Ser Phe Leu Thr Arg Tyr Ala Val Tyr Asn Ser Ile Leu
355 360 365
Arg Lys Leu Asn His Ala Lys Met Phe Ala Thr Phe Thr Leu Pro Asp
370 375 380
Ala Thr Ala His Pro Ile Trp Thr Arg Phe Asp Lys Leu Gly Gly Asn
385 390 395 400
Leu His Gln Tyr Thr Phe Leu Phe Asn Glu Phe Gly Glu Arg Arg His
405 410 415
Ala Ile Arg Phe His Lys Leu Leu Lys Val Glu Asn Gly Val Ala Arg
420 425 430
Glu Val Asp Asp Val Thr Val Pro Ile Ser Met Ser Glu Gln Leu Asp
435 440 445
Asn Leu Leu Pro Arg Asp Pro Asn Glu Pro Ile Ala Leu Tyr Phe Arg
450 455 460
Asp Tyr Gly Ala Glu Gln His Phe Thr Gly Glu Phe Gly Gly Ala Lys
465 470 475 480
Ile Gln Cys Arg Arg Asp Gln Leu Ala His Met His Arg Arg Arg Gly
485 490 495
Ala Arg Asp Val Tyr Leu Asn Val Ser Val Arg Val Gln Ser Gln Ser
500 505 510
Glu Ala Arg Gly Glu Arg Arg Pro Pro Tyr Ala Ala Val Phe Arg Leu
515 520 525
Val Gly Asp Asn His Arg Ala Phe Val His Phe Asp Lys Leu Ser Asp
530 535 540
Tyr Leu Ala Glu His Pro Asp Asp Gly Lys Leu Gly Ser Glu Gly Leu
545 550 555 560
Leu Ser Gly Leu Arg Val Met Ser Val Asp Leu Gly Leu Arg Thr Ser
565 570 575
Ala Ser Ile Ser Val Phe Arg Val Ala Arg Lys Asp Glu Leu Lys Pro
580 585 590
Asn Ser Lys Gly Arg Val Pro Phe Phe Phe Pro Ile Lys Gly Asn Asp
595 600 605
Asn Leu Val Ala Val His Glu Arg Ser Gln Leu Leu Lys Leu Pro Gly
610 615 620
Glu Thr Glu Ser Lys Asp Leu Arg Ala Ile Arg Glu Glu Arg Gln Arg
625 630 635 640
Thr Leu Arg Gln Leu Arg Thr Gln Leu Ala Tyr Leu Arg Leu Leu Val
645 650 655
Arg Cys Gly Ser Glu Asp Val Gly Arg Arg Glu Arg Ser Trp Ala Lys
660 665 670
Leu Ile Glu Gln Pro Val Asp Ala Ala Asn His Met Thr Pro Asp Trp
675 680 685
Arg Glu Ala Phe Glu Asn Glu Leu Gln Lys Leu Lys Ser Leu His Gly
690 695 700
Ile Cys Ser Asp Lys Glu Trp Met Asp Ala Val Tyr Glu Ser Val Arg
705 710 715 720
Arg Val Trp Arg His Met Gly Lys Gln Val Arg Asp Trp Arg Lys Asp
725 730 735
Val Arg Ser Gly Glu Arg Pro Lys Ile Arg Gly Tyr Ala Lys Asp Val
740 745 750
Val Gly Gly Asn Ser Ile Glu Gln Ile Glu Tyr Leu Glu Arg Gln Tyr
755 760 765
Lys Phe Leu Lys Ser Trp Ser Phe Phe Gly Lys Val Ser Gly Gln Val
770 775 780
Ile Arg Ala Glu Lys Gly Ser Arg Phe Ala Ile Thr Leu Arg Glu His
785 790 795 800
Ile Asp His Ala Lys Glu Asp Arg Leu Lys Lys Leu Ala Asp Arg Ile
805 810 815
Ile Met Glu Ala Leu Gly Tyr Val Tyr Ala Leu Asp Glu Arg Gly Lys
820 825 830
Gly Lys Trp Val Ala Lys Tyr Pro Pro Cys Gln Leu Ile Leu Leu Glu
835 840 845
Glu Leu Ser Glu Tyr Gln Phe Asn Asn Asp Arg Pro Pro Ser Glu Asn
850 855 860
Asn Gln Leu Met Gln Trp Ser His Arg Gly Val Phe Gln Glu Leu Ile
865 870 875 880
Asn Gln Ala Gln Val His Asp Leu Leu Val Gly Thr Met Tyr Ala Ala
885 890 895
Phe Ser Ser Arg Phe Asp Ala Arg Thr Gly Ala Pro Gly Ile Arg Cys
900 905 910
Arg Arg Val Pro Ala Arg Cys Thr Gln Glu His Asn Pro Glu Pro Phe
915 920 925
Pro Trp Trp Leu Asn Lys Phe Val Val Glu His Thr Leu Asp Ala Cys
930 935 940
Pro Leu Arg Ala Asp Asp Leu Ile Pro Thr Gly Glu Gly Glu Ile Phe
945 950 955 960
Val Ser Pro Phe Ser Ala Glu Glu Gly Asp Phe His Gln Ile His Ala
965 970 975
Asp Leu Asn Ala Ala Gln Asn Leu Gln Gln Arg Leu Trp Ser Asp Phe
980 985 990
Asp Ile Ser Gln Ile Arg Leu Arg Cys Asp Trp Gly Glu Val Asp Gly
995 1000 1005
Glu Leu Val Leu Ile Pro Arg Leu Thr Gly Lys Arg Thr Ala Asp
1010 1015 1020
Ser Tyr Ser Asn Lys Val Phe Tyr Thr Asn Thr Gly Val Thr Tyr
1025 1030 1035
Tyr Glu Arg Glu Arg Gly Lys Lys Arg Arg Lys Val Phe Ala Gln
1040 1045 1050
Glu Lys Leu Ser Glu Glu Glu Ala Glu Leu Leu Val Glu Ala Asp
1055 1060 1065
Glu Ala Arg Glu Lys Ser Val Val Leu Met Arg Asp Pro Ser Gly
1070 1075 1080
Ile Ile Asn Arg Gly Asn Trp Thr Arg Gln Lys Glu Phe Trp Ser
1085 1090 1095
Met Val Asn Gln Arg Ile Glu Gly Tyr Leu Val Lys Gln Ile Arg
1100 1105 1110
Ser Arg Val Pro Leu Gln Asp Ser Ala Cys Glu Asn Thr Gly Asp
1115 1120 1125
Ile
<210> 549
<211> 872
<212> PRT
<213> Alicyclobacillus contaminans
<400> 549
Met Gly Phe Asn Thr Ala Glu Leu Leu Arg Lys Val Glu Glu Glu Met
1 5 10 15
Arg Lys Thr Ser Val Gly Phe Asp Thr Asp Asn Pro Phe Ala His Arg
20 25 30
Ile Thr Arg Arg Ala Ile Arg Gly Trp Asp Arg Ile Ala Glu Ala Trp
35 40 45
Arg Arg Leu Pro Pro Asp Ala Pro Glu Ser Glu Tyr Ile Glu Ala Phe
50 55 60
Lys Asp Ile Gln Arg Lys Asn Pro Arg Lys Ile Gly Ser Glu Pro Leu
65 70 75 80
Phe Lys Asn Leu Ala Ala Pro Gly Val Arg Ser Glu Leu Leu Asn Asn
85 90 95
Pro Gln Val Leu Ile Thr Phe Ala Lys Tyr Asn Glu Leu Gln Arg Gln
100 105 110
Leu Ala Lys Ala Lys Gln Phe Ala Gln Lys Thr Leu Pro His Pro Val
115 120 125
Phe His Pro Val Trp Val Arg Tyr Asp Lys Leu Gly Gly Asn Leu His
130 135 140
His Tyr Gln Ile Glu Pro Ala Val His Ala Asn Asp Thr His Lys Val
145 150 155 160
Lys Phe Ser Ser Leu Leu Leu Pro Gln Glu Asp Gly Ser Tyr Ala Glu
165 170 175
Val Lys Asp Val Thr Val Ser Leu Ala Pro Ser Leu Gln Phe Pro Thr
180 185 190
Gly Leu Val His Pro Lys Val Thr Thr Pro Pro Arg Thr Gly Leu Val
195 200 205
Thr Val Met Asp Glu Glu Ala Gly Lys Pro Val Val Cys Tyr Arg Asp
210 215 220
Arg Gly His Asp Ala Leu Val Pro Val Ala Phe Gly Gly Ala Lys Leu
225 230 235 240
Gln Phe Asn Arg Ala His Leu Ser Ala Gly Tyr Arg Lys Gly Val Leu
245 250 255
Ser Ala Gly Gly Gly Gly Ser Ile Tyr Phe Asn Val Thr Leu Asp Val
260 265 270
Gln Val Pro Asn Glu Arg Asp Val Ser Lys Thr Phe Ser Phe Ser Arg
275 280 285
Asp Arg Asp Leu Val Ser Leu Lys Ala Glu Glu Leu Lys Arg Tyr Met
290 295 300
Glu Thr Lys Pro Leu Gly Met Pro Gly Val Arg Val Met Ser Val Asp
305 310 315 320
Leu Gly Val Arg Tyr Gly Ala Ala Ile Ser Val Phe Glu Val Lys Pro
325 330 335
Phe Ala Glu Val Arg Lys Asp Lys Leu His Tyr Pro Ile Thr Gly Cys
340 345 350
Glu Gly Phe Val Ala Glu His Glu Arg Ser Val Ile Leu Lys Leu Pro
355 360 365
Gly Glu Gly Val Arg Thr Ala Gly Lys Gln Ser Glu Arg Lys Gln Ala
370 375 380
Leu Ala Ala Ile Arg Ala Glu Met Ser Ile Leu Arg Lys Trp Leu Arg
385 390 395 400
Val Ser Gln Val Thr Glu Glu Asp Arg Ala Lys Ala Val Arg Gly Leu
405 410 415
Leu Glu Asp Glu Arg Gly Gly Gly Trp Thr Met Asp Pro Gly Glu Asp
420 425 430
Ser Asp His Gln Pro Leu Gln Gln Phe Leu His Glu Ala Arg Leu Ala
435 440 445
Val Gly Glu Leu Val Asn Leu Val His Leu Ser Pro Ala Glu Trp Glu
450 455 460
Arg Ala Val Ile Glu Arg His Arg Arg Leu Glu Arg Ile Thr Ala Ser
465 470 475 480
His Ile Arg Val Phe Gln Thr Met Arg Lys Val Trp Gly Lys Arg Arg
485 490 495
Asn Glu Asp Ala Ala His Thr Gly Gly Ile Ser Leu Ala His Ile Glu
500 505 510
His Leu Ile Gln Gln Arg Lys Leu Phe Ile Arg Trp Ser Thr His Ala
515 520 525
Arg Thr Tyr Gly Glu Val Arg Arg Leu Pro Lys His Glu Gly Phe Ala
530 535 540
Lys Arg Leu Gln Lys His Thr Asn His Val Lys Glu Asp Arg Ile Lys
545 550 555 560
Lys Leu Ala Asp Met Ile Val Met Ala Ala Arg Gly Tyr Arg Phe Leu
565 570 575
Asp Lys Arg Ala Arg Trp Val Lys Thr Arg His Ala Pro Cys Asp Leu
580 585 590
Ile Leu Phe Glu Asp Leu Ser Arg Tyr Arg Phe Thr Met Asp Arg Pro
595 600 605
Pro Thr Glu Asn Ser Gln Leu Met Asn Trp Ser His Arg Glu Leu Leu
610 615 620
Lys Thr Val Lys Met Gln Ala Ala Leu Phe Gly Ile Gly Val Gly Thr
625 630 635 640
Val Pro Ala Ala Phe Thr Ser Arg Phe Asp Ala Gln Thr Gly Ala Pro
645 650 655
Gly Leu Arg Cys Lys Arg Val Thr Lys Gln Asp Lys Glu Lys Thr Pro
660 665 670
Phe Trp Leu Ile Gln Phe Ala Glu Ile Thr Gly Val Asn Val Thr Asn
675 680 685
Val Glu Pro Gly Gln Leu Ile Pro Val Asp Gly Gly Glu Trp Phe Val
690 695 700
Ser Pro Lys Gly Pro Arg Ala Ala Asp Gly Leu Lys Cys Val His Ala
705 710 715 720
Asp Ile Asn Ala Ala His Asn Leu Gln Arg Arg Phe Trp Ile Pro Arg
725 730 735
Leu Pro Ser Val Lys Cys Arg Arg Tyr Val Glu Ala Glu Gly Phe Ala
740 745 750
Ala Val Pro Ser Ser Thr Ala Phe Met Lys Val His Gly Lys Gly Ala
755 760 765
Phe Val Ser Val Asp Gly Glu Phe Tyr Glu Tyr Gln Lys Gly Arg Arg
770 775 780
Val Ala Val Asn Arg Ala Asp Arg Thr Ser Ser Thr Leu Asp Glu Asp
785 790 795 800
Glu Gly Asp Ile Gly Glu Glu Met Leu Val Ser Ser Asn Gly Ala Gly
805 810 815
Glu Phe Val Arg Met Phe Tyr Asp Glu Ser Gly Tyr Val Gly Tyr Gly
820 825 830
Arg Trp Met Asp Ser Lys Val Phe Trp Gly Lys Val Arg Gln Ile Val
835 840 845
His Arg Ala Ile Gln Asp Gln Val Glu Lys Arg Ala Ala Ala Arg Gly
850 855 860
Glu Asn Gly Ala Thr Ser Ser Arg
865 870
<210> 550
<211> 1149
<212> PRT
<213> Desulfovibrio inopinatus
<400> 550
Met Pro Thr Arg Thr Ile Asn Leu Lys Leu Val Leu Gly Lys Asn Pro
1 5 10 15
Glu Asn Ala Thr Leu Arg Arg Ala Leu Phe Ser Thr His Arg Leu Val
20 25 30
Asn Gln Ala Thr Lys Arg Ile Glu Glu Phe Leu Leu Leu Cys Arg Gly
35 40 45
Glu Ala Tyr Arg Thr Val Asp Asn Glu Gly Lys Glu Ala Glu Ile Pro
50 55 60
Arg His Ala Val Gln Glu Glu Ala Leu Ala Phe Ala Lys Ala Ala Gln
65 70 75 80
Arg His Asn Gly Cys Ile Ser Thr Tyr Glu Asp Gln Glu Ile Leu Asp
85 90 95
Val Leu Arg Gln Leu Tyr Glu Arg Leu Val Pro Ser Val Asn Glu Asn
100 105 110
Asn Glu Ala Gly Asp Ala Gln Ala Ala Asn Ala Trp Val Ser Pro Leu
115 120 125
Met Ser Ala Glu Ser Glu Gly Gly Leu Ser Val Tyr Asp Lys Val Leu
130 135 140
Asp Pro Pro Pro Val Trp Met Lys Leu Lys Glu Glu Lys Ala Pro Gly
145 150 155 160
Trp Glu Ala Ala Ser Gln Ile Trp Ile Gln Ser Asp Glu Gly Gln Ser
165 170 175
Leu Leu Asn Lys Pro Gly Ser Pro Pro Arg Trp Ile Arg Lys Leu Arg
180 185 190
Ser Gly Gln Pro Trp Gln Asp Asp Phe Val Ser Asp Gln Lys Lys Lys
195 200 205
Gln Asp Glu Leu Thr Lys Gly Asn Ala Pro Leu Ile Lys Gln Leu Lys
210 215 220
Glu Met Gly Leu Leu Pro Leu Val Asn Pro Phe Phe Arg His Leu Leu
225 230 235 240
Asp Pro Glu Gly Lys Gly Val Ser Pro Trp Asp Arg Leu Ala Val Arg
245 250 255
Ala Ala Val Ala His Phe Ile Ser Trp Glu Ser Trp Asn His Arg Thr
260 265 270
Arg Ala Glu Tyr Asn Ser Leu Lys Leu Arg Arg Asp Glu Phe Glu Ala
275 280 285
Ala Ser Asp Glu Phe Lys Asp Asp Phe Thr Leu Leu Arg Gln Tyr Glu
290 295 300
Ala Lys Arg His Ser Thr Leu Lys Ser Ile Ala Leu Ala Asp Asp Ser
305 310 315 320
Asn Pro Tyr Arg Ile Gly Val Arg Ser Leu Arg Ala Trp Asn Arg Val
325 330 335
Arg Glu Glu Trp Ile Asp Lys Gly Ala Thr Glu Glu Gln Arg Val Thr
340 345 350
Ile Leu Ser Lys Leu Gln Thr Gln Leu Arg Gly Lys Phe Gly Asp Pro
355 360 365
Asp Leu Phe Asn Trp Leu Ala Gln Asp Arg His Val His Leu Trp Ser
370 375 380
Pro Arg Asp Ser Val Thr Pro Leu Val Arg Ile Asn Ala Val Asp Lys
385 390 395 400
Val Leu Arg Arg Arg Lys Pro Tyr Ala Leu Met Thr Phe Ala His Pro
405 410 415
Arg Phe His Pro Arg Trp Ile Leu Tyr Glu Ala Pro Gly Gly Ser Asn
420 425 430
Leu Arg Gln Tyr Ala Leu Asp Cys Thr Glu Asn Ala Leu His Ile Thr
435 440 445
Leu Pro Leu Leu Val Asp Asp Ala His Gly Thr Trp Ile Glu Lys Lys
450 455 460
Ile Arg Val Pro Leu Ala Pro Ser Gly Gln Ile Gln Asp Leu Thr Leu
465 470 475 480
Glu Lys Leu Glu Lys Lys Lys Asn Arg Leu Tyr Tyr Arg Ser Gly Phe
485 490 495
Gln Gln Phe Ala Gly Leu Ala Gly Gly Ala Glu Val Leu Phe His Arg
500 505 510
Pro Tyr Met Glu His Asp Glu Arg Ser Glu Glu Ser Leu Leu Glu Arg
515 520 525
Pro Gly Ala Val Trp Phe Lys Leu Thr Leu Asp Val Ala Thr Gln Ala
530 535 540
Pro Pro Asn Trp Leu Asp Gly Lys Gly Arg Val Arg Thr Pro Pro Glu
545 550 555 560
Val His His Phe Lys Thr Ala Leu Ser Asn Lys Ser Lys His Thr Arg
565 570 575
Thr Leu Gln Pro Gly Leu Arg Val Leu Ser Val Asp Leu Gly Met Arg
580 585 590
Thr Phe Ala Ser Cys Ser Val Phe Glu Leu Ile Glu Gly Lys Pro Glu
595 600 605
Thr Gly Arg Ala Phe Pro Val Ala Asp Glu Arg Ser Met Asp Ser Pro
610 615 620
Asn Lys Leu Trp Ala Lys His Glu Arg Ser Phe Lys Leu Thr Leu Pro
625 630 635 640
Gly Glu Thr Pro Ser Arg Lys Glu Glu Glu Glu Arg Ser Ile Ala Arg
645 650 655
Ala Glu Ile Tyr Ala Leu Lys Arg Asp Ile Gln Arg Leu Lys Ser Leu
660 665 670
Leu Arg Leu Gly Glu Glu Asp Asn Asp Asn Arg Arg Asp Ala Leu Leu
675 680 685
Glu Gln Phe Phe Lys Gly Trp Gly Glu Glu Asp Val Val Pro Gly Gln
690 695 700
Ala Phe Pro Arg Ser Leu Phe Gln Gly Leu Gly Ala Ala Pro Phe Arg
705 710 715 720
Ser Thr Pro Glu Leu Trp Arg Gln His Cys Gln Thr Tyr Tyr Asp Lys
725 730 735
Ala Glu Ala Cys Leu Ala Lys His Ile Ser Asp Trp Arg Lys Arg Thr
740 745 750
Arg Pro Arg Pro Thr Ser Arg Glu Met Trp Tyr Lys Thr Arg Ser Tyr
755 760 765
His Gly Gly Lys Ser Ile Trp Met Leu Glu Tyr Leu Asp Ala Val Arg
770 775 780
Lys Leu Leu Leu Ser Trp Ser Leu Arg Gly Arg Thr Tyr Gly Ala Ile
785 790 795 800
Asn Arg Gln Asp Thr Ala Arg Phe Gly Ser Leu Ala Ser Arg Leu Leu
805 810 815
His His Ile Asn Ser Leu Lys Glu Asp Arg Ile Lys Thr Gly Ala Asp
820 825 830
Ser Ile Val Gln Ala Ala Arg Gly Tyr Ile Pro Leu Pro His Gly Lys
835 840 845
Gly Trp Glu Gln Arg Tyr Glu Pro Cys Gln Leu Ile Leu Phe Glu Asp
850 855 860
Leu Ala Arg Tyr Arg Phe Arg Val Asp Arg Pro Arg Arg Glu Asn Ser
865 870 875 880
Gln Leu Met Gln Trp Asn His Arg Ala Ile Val Ala Glu Thr Thr Met
885 890 895
Gln Ala Glu Leu Tyr Gly Gln Ile Val Glu Asn Thr Ala Ala Gly Phe
900 905 910
Ser Ser Arg Phe His Ala Ala Thr Gly Ala Pro Gly Val Arg Cys Arg
915 920 925
Phe Leu Leu Glu Arg Asp Phe Asp Asn Asp Leu Pro Lys Pro Tyr Leu
930 935 940
Leu Arg Glu Leu Ser Trp Met Leu Gly Asn Thr Lys Val Glu Ser Glu
945 950 955 960
Glu Glu Lys Leu Arg Leu Leu Ser Glu Lys Ile Arg Pro Gly Ser Leu
965 970 975
Val Pro Trp Asp Gly Gly Glu Gln Phe Ala Thr Leu His Pro Lys Arg
980 985 990
Gln Thr Leu Cys Val Ile His Ala Asp Met Asn Ala Ala Gln Asn Leu
995 1000 1005
Gln Arg Arg Phe Phe Gly Arg Cys Gly Glu Ala Phe Arg Leu Val
1010 1015 1020
Cys Gln Pro His Gly Asp Asp Val Leu Arg Leu Ala Ser Thr Pro
1025 1030 1035
Gly Ala Arg Leu Leu Gly Ala Leu Gln Gln Leu Glu Asn Gly Gln
1040 1045 1050
Gly Ala Phe Glu Leu Val Arg Asp Met Gly Ser Thr Ser Gln Met
1055 1060 1065
Asn Arg Phe Val Met Lys Ser Leu Gly Lys Lys Lys Ile Lys Pro
1070 1075 1080
Leu Gln Asp Asn Asn Gly Asp Asp Glu Leu Glu Asp Val Leu Ser
1085 1090 1095
Val Leu Pro Glu Glu Asp Asp Thr Gly Arg Ile Thr Val Phe Arg
1100 1105 1110
Asp Ser Ser Gly Ile Phe Phe Pro Cys Asn Val Trp Ile Pro Ala
1115 1120 1125
Lys Gln Phe Trp Pro Ala Val Arg Ala Met Ile Trp Lys Val Met
1130 1135 1140
Ala Ser His Ser Leu Gly
1145
<210> 551
<211> 1194
<212> PRT
<213> Desulfonatronum thiodismutans
<400> 551
Met Val Leu Gly Arg Lys Asp Asp Thr Ala Glu Leu Arg Arg Ala Leu
1 5 10 15
Trp Thr Thr His Glu His Val Asn Leu Ala Val Ala Glu Val Glu Arg
20 25 30
Val Leu Leu Arg Cys Arg Gly Arg Ser Tyr Trp Thr Leu Asp Arg Arg
35 40 45
Gly Asp Pro Val His Val Pro Glu Ser Gln Val Ala Glu Asp Ala Leu
50 55 60
Ala Met Ala Arg Glu Ala Gln Arg Arg Asn Gly Trp Pro Val Val Gly
65 70 75 80
Glu Asp Glu Glu Ile Leu Leu Ala Leu Arg Tyr Leu Tyr Glu Gln Ile
85 90 95
Val Pro Ser Cys Leu Leu Asp Asp Leu Gly Lys Pro Leu Lys Gly Asp
100 105 110
Ala Gln Lys Ile Gly Thr Asn Tyr Ala Gly Pro Leu Phe Asp Ser Asp
115 120 125
Thr Cys Arg Arg Asp Glu Gly Lys Asp Val Ala Cys Cys Gly Pro Phe
130 135 140
His Glu Val Ala Gly Lys Tyr Leu Gly Ala Leu Pro Glu Trp Ala Thr
145 150 155 160
Pro Ile Ser Lys Gln Glu Phe Asp Gly Lys Asp Ala Ser His Leu Arg
165 170 175
Phe Lys Ala Thr Gly Gly Asp Asp Ala Phe Phe Arg Val Ser Ile Glu
180 185 190
Lys Ala Asn Ala Trp Tyr Glu Asp Pro Ala Asn Gln Asp Ala Leu Lys
195 200 205
Asn Lys Ala Tyr Asn Lys Asp Asp Trp Lys Lys Glu Lys Asp Lys Gly
210 215 220
Ile Ser Ser Trp Ala Val Lys Tyr Ile Gln Lys Gln Leu Gln Leu Gly
225 230 235 240
Gln Asp Pro Arg Thr Glu Val Arg Arg Lys Leu Trp Leu Glu Leu Gly
245 250 255
Leu Leu Pro Leu Phe Ile Pro Val Phe Asp Lys Thr Met Val Gly Asn
260 265 270
Leu Trp Asn Arg Leu Ala Val Arg Leu Ala Leu Ala His Leu Leu Ser
275 280 285
Trp Glu Ser Trp Asn His Arg Ala Val Gln Asp Gln Ala Leu Ala Arg
290 295 300
Ala Lys Arg Asp Glu Leu Ala Ala Leu Phe Leu Gly Met Glu Asp Gly
305 310 315 320
Phe Ala Gly Leu Arg Glu Tyr Glu Leu Arg Arg Asn Glu Ser Ile Lys
325 330 335
Gln His Ala Phe Glu Pro Val Asp Arg Pro Tyr Val Val Ser Gly Arg
340 345 350
Ala Leu Arg Ser Trp Thr Arg Val Arg Glu Glu Trp Leu Arg His Gly
355 360 365
Asp Thr Gln Glu Ser Arg Lys Asn Ile Cys Asn Arg Leu Gln Asp Arg
370 375 380
Leu Arg Gly Lys Phe Gly Asp Pro Asp Val Phe His Trp Leu Ala Glu
385 390 395 400
Asp Gly Gln Glu Ala Leu Trp Lys Glu Arg Asp Cys Val Thr Ser Phe
405 410 415
Ser Leu Leu Asn Asp Ala Asp Gly Leu Leu Glu Lys Arg Lys Gly Tyr
420 425 430
Ala Leu Met Thr Phe Ala Asp Ala Arg Leu His Pro Arg Trp Ala Met
435 440 445
Tyr Glu Ala Pro Gly Gly Ser Asn Leu Arg Thr Tyr Gln Ile Arg Lys
450 455 460
Thr Glu Asn Gly Leu Trp Ala Asp Val Val Leu Leu Ser Pro Arg Asn
465 470 475 480
Glu Ser Ala Ala Val Glu Glu Lys Thr Phe Asn Val Arg Leu Ala Pro
485 490 495
Ser Gly Gln Leu Ser Asn Val Ser Phe Asp Gln Ile Gln Lys Gly Ser
500 505 510
Lys Met Val Gly Arg Cys Arg Tyr Gln Ser Ala Asn Gln Gln Phe Glu
515 520 525
Gly Leu Leu Gly Gly Ala Glu Ile Leu Phe Asp Arg Lys Arg Ile Ala
530 535 540
Asn Glu Gln His Gly Ala Thr Asp Leu Ala Ser Lys Pro Gly His Val
545 550 555 560
Trp Phe Lys Leu Thr Leu Asp Val Arg Pro Gln Ala Pro Gln Gly Trp
565 570 575
Leu Asp Gly Lys Gly Arg Pro Ala Leu Pro Pro Glu Ala Lys His Phe
580 585 590
Lys Thr Ala Leu Ser Asn Lys Ser Lys Phe Ala Asp Gln Val Arg Pro
595 600 605
Gly Leu Arg Val Leu Ser Val Asp Leu Gly Val Arg Ser Phe Ala Ala
610 615 620
Cys Ser Val Phe Glu Leu Val Arg Gly Gly Pro Asp Gln Gly Thr Tyr
625 630 635 640
Phe Pro Ala Ala Asp Gly Arg Thr Val Asp Asp Pro Glu Lys Leu Trp
645 650 655
Ala Lys His Glu Arg Ser Phe Lys Ile Thr Leu Pro Gly Glu Asn Pro
660 665 670
Ser Arg Lys Glu Glu Ile Ala Arg Arg Ala Ala Met Glu Glu Leu Arg
675 680 685
Ser Leu Asn Gly Asp Ile Arg Arg Leu Lys Ala Ile Leu Arg Leu Ser
690 695 700
Val Leu Gln Glu Asp Asp Pro Arg Thr Glu His Leu Arg Leu Phe Met
705 710 715 720
Glu Ala Ile Val Asp Asp Pro Ala Lys Ser Ala Leu Asn Ala Glu Leu
725 730 735
Phe Lys Gly Phe Gly Asp Asp Arg Phe Arg Ser Thr Pro Asp Leu Trp
740 745 750
Lys Gln His Cys His Phe Phe His Asp Lys Ala Glu Lys Val Val Ala
755 760 765
Glu Arg Phe Ser Arg Trp Arg Thr Glu Thr Arg Pro Lys Ser Ser Ser
770 775 780
Trp Gln Asp Trp Arg Glu Arg Arg Gly Tyr Ala Gly Gly Lys Ser Tyr
785 790 795 800
Trp Ala Val Thr Tyr Leu Glu Ala Val Arg Gly Leu Ile Leu Arg Trp
805 810 815
Asn Met Arg Gly Arg Thr Tyr Gly Glu Val Asn Arg Gln Asp Lys Lys
820 825 830
Gln Phe Gly Thr Val Ala Ser Ala Leu Leu His His Ile Asn Gln Leu
835 840 845
Lys Glu Asp Arg Ile Lys Thr Gly Ala Asp Met Ile Ile Gln Ala Ala
850 855 860
Arg Gly Phe Val Pro Arg Lys Asn Gly Ala Gly Trp Val Gln Val His
865 870 875 880
Glu Pro Cys Arg Leu Ile Leu Phe Glu Asp Leu Ala Arg Tyr Arg Phe
885 890 895
Arg Thr Asp Arg Ser Arg Arg Glu Asn Ser Arg Leu Met Arg Trp Ser
900 905 910
His Arg Glu Ile Val Asn Glu Val Gly Met Gln Gly Glu Leu Tyr Gly
915 920 925
Leu His Val Asp Thr Thr Glu Ala Gly Phe Ser Ser Arg Tyr Leu Ala
930 935 940
Ser Ser Gly Ala Pro Gly Val Arg Cys Arg His Leu Val Glu Glu Asp
945 950 955 960
Phe His Asp Gly Leu Pro Gly Met His Leu Val Gly Glu Leu Asp Trp
965 970 975
Leu Leu Pro Lys Asp Lys Asp Arg Thr Ala Asn Glu Ala Arg Arg Leu
980 985 990
Leu Gly Gly Met Val Arg Pro Gly Met Leu Val Pro Trp Asp Gly Gly
995 1000 1005
Glu Leu Phe Ala Thr Leu Asn Ala Ala Ser Gln Leu His Val Ile
1010 1015 1020
His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln Arg Arg Phe Trp
1025 1030 1035
Gly Arg Cys Gly Glu Ala Ile Arg Ile Val Cys Asn Gln Leu Ser
1040 1045 1050
Val Asp Gly Ser Thr Arg Tyr Glu Met Ala Lys Ala Pro Lys Ala
1055 1060 1065
Arg Leu Leu Gly Ala Leu Gln Gln Leu Lys Asn Gly Asp Ala Pro
1070 1075 1080
Phe His Leu Thr Ser Ile Pro Asn Ser Gln Lys Pro Glu Asn Ser
1085 1090 1095
Tyr Val Met Thr Pro Thr Asn Ala Gly Lys Lys Tyr Arg Ala Gly
1100 1105 1110
Pro Gly Glu Lys Ser Ser Gly Glu Glu Asp Glu Leu Ala Leu Asp
1115 1120 1125
Ile Val Glu Gln Ala Glu Glu Leu Ala Gln Gly Arg Lys Thr Phe
1130 1135 1140
Phe Arg Asp Pro Ser Gly Val Phe Phe Ala Pro Asp Arg Trp Leu
1145 1150 1155
Pro Ser Glu Ile Tyr Trp Ser Arg Ile Arg Arg Arg Ile Trp Gln
1160 1165 1170
Val Thr Leu Glu Arg Asn Ser Ser Gly Arg Gln Glu Arg Ala Glu
1175 1180 1185
Met Asp Glu Met Pro Tyr
1190
<210> 552
<211> 1388
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(1388)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 552
Met Ser Leu Asn Arg Ile Tyr Gln Gly Arg Val Ala Ala Val Glu Thr
1 5 10 15
Gly Thr Ala Leu Ala Lys Gly Asn Val Glu Trp Met Pro Ala Ala Gly
20 25 30
Gly Asp Glu Val Leu Trp Gln His His Glu Leu Phe Gln Ala Ala Ile
35 40 45
Asn Tyr Tyr Leu Val Ala Leu Leu Ala Leu Ala Asp Lys Asn Asn Pro
50 55 60
Val Leu Gly Pro Leu Ile Ser Gln Met Asp Asn Pro Gln Ser Pro Tyr
65 70 75 80
His Val Trp Gly Ser Phe Arg Arg Gln Gly Arg Gln Arg Thr Gly Leu
85 90 95
Ser Gln Ala Val Ala Pro Tyr Ile Thr Pro Gly Asn Asn Ala Pro Thr
100 105 110
Leu Asp Glu Val Phe Arg Ser Ile Leu Ala Gly Asn Pro Thr Asp Arg
115 120 125
Ala Thr Leu Asp Ala Ala Leu Met Gln Leu Leu Lys Ala Cys Asp Gly
130 135 140
Ala Gly Ala Ile Gln Gln Glu Gly Arg Ser Tyr Trp Pro Lys Phe Cys
145 150 155 160
Asp Pro Asp Ser Thr Ala Asn Phe Ala Gly Asp Pro Ala Met Leu Arg
165 170 175
Arg Glu Gln His Arg Leu Leu Leu Pro Gln Val Leu His Asp Pro Ala
180 185 190
Ile Thr His Asp Ser Pro Ala Leu Gly Ser Phe Asp Thr Tyr Ser Ile
195 200 205
Ala Thr Pro Asp Thr Arg Thr Pro Gln Leu Thr Gly Pro Lys Ala Arg
210 215 220
Ala Arg Leu Glu Gln Ala Ile Thr Leu Trp Arg Val Arg Leu Pro Glu
225 230 235 240
Ser Ala Ala Asp Phe Asp Arg Leu Ala Ser Ser Leu Lys Lys Ile Pro
245 250 255
Asp Asp Asp Ser Arg Leu Asn Leu Gln Gly Tyr Val Gly Ser Ser Ala
260 265 270
Lys Gly Glu Val Gln Ala Arg Leu Phe Ala Leu Leu Leu Phe Arg His
275 280 285
Leu Glu Arg Ser Ser Phe Thr Leu Gly Leu Leu Arg Ser Ala Thr Pro
290 295 300
Pro Pro Lys Asn Ala Glu Thr Pro Pro Pro Ala Gly Val Pro Leu Pro
305 310 315 320
Ala Ala Ser Ala Ala Asp Pro Val Arg Ile Ala Arg Gly Lys Arg Ser
325 330 335
Phe Val Phe Arg Ala Phe Thr Ser Leu Pro Cys Trp His Gly Gly Asp
340 345 350
Asn Ile His Pro Thr Trp Lys Ser Phe Asp Ile Ala Ala Phe Lys Tyr
355 360 365
Ala Leu Thr Val Ile Asn Gln Ile Glu Glu Lys Thr Lys Glu Arg Gln
370 375 380
Lys Glu Cys Ala Glu Leu Glu Thr Asp Phe Asp Tyr Met His Gly Arg
385 390 395 400
Leu Ala Lys Ile Pro Val Lys Tyr Thr Thr Gly Glu Ala Glu Pro Pro
405 410 415
Pro Ile Leu Ala Asn Asp Leu Arg Ile Pro Leu Leu Arg Glu Leu Leu
420 425 430
Gln Asn Ile Lys Val Asp Thr Ala Leu Thr Asp Gly Glu Ala Val Ser
435 440 445
Tyr Gly Leu Gln Arg Arg Thr Ile Arg Gly Phe Arg Glu Leu Arg Arg
450 455 460
Ile Trp Arg Gly His Ala Pro Ala Gly Thr Val Phe Ser Ser Glu Leu
465 470 475 480
Lys Glu Lys Leu Ala Gly Glu Leu Arg Gln Phe Gln Thr Asp Asn Ser
485 490 495
Thr Thr Ile Gly Ser Val Gln Leu Phe Asn Glu Leu Ile Gln Asn Pro
500 505 510
Lys Tyr Trp Pro Ile Trp Gln Ala Pro Asp Val Glu Thr Ala Arg Gln
515 520 525
Trp Ala Asp Ala Gly Phe Ala Asp Asp Pro Leu Ala Ala Leu Val Gln
530 535 540
Glu Ala Glu Leu Gln Glu Asp Ile Asp Ala Leu Lys Ala Pro Val Lys
545 550 555 560
Leu Thr Pro Ala Asp Pro Glu Tyr Ser Arg Arg Gln Tyr Asp Phe Asn
565 570 575
Ala Val Ser Lys Phe Gly Ala Gly Ser Arg Ser Ala Asn Arg His Glu
580 585 590
Pro Gly Gln Thr Glu Arg Gly His Asn Thr Phe Thr Thr Glu Ile Ala
595 600 605
Ala Arg Asn Ala Ala Asp Gly Asn Arg Trp Arg Ala Thr His Val Arg
610 615 620
Ile His Tyr Ser Ala Pro Arg Leu Leu Arg Asp Gly Leu Arg Arg Pro
625 630 635 640
Asp Thr Asp Gly Asn Glu Ala Leu Glu Ala Val Pro Trp Leu Gln Pro
645 650 655
Met Met Glu Ala Leu Ala Pro Leu Pro Thr Leu Pro Gln Asp Leu Thr
660 665 670
Gly Met Pro Val Phe Leu Met Pro Asp Val Thr Leu Ser Gly Glu Arg
675 680 685
Arg Ile Leu Leu Asn Leu Pro Val Thr Leu Glu Pro Ala Ala Leu Val
690 695 700
Glu Gln Leu Gly Asn Ala Gly Arg Trp Gln Asn Gln Phe Phe Gly Ser
705 710 715 720
Arg Glu Asp Pro Phe Ala Leu Arg Trp Pro Ala Asp Gly Ala Val Lys
725 730 735
Thr Ala Lys Gly Lys Thr His Ile Pro Trp His Gln Asp Arg Asp His
740 745 750
Phe Thr Val Leu Gly Val Asp Leu Gly Thr Arg Asp Ala Gly Ala Leu
755 760 765
Ala Leu Leu Asn Val Thr Ala Gln Lys Pro Ala Lys Pro Val His Arg
770 775 780
Ile Ile Gly Glu Ala Asp Gly Arg Thr Trp Tyr Ala Ser Leu Ala Asp
785 790 795 800
Ala Arg Met Ile Arg Leu Pro Gly Glu Asp Ala Arg Leu Phe Val Arg
805 810 815
Gly Lys Leu Val Gln Glu Pro Tyr Gly Glu Arg Gly Arg Asn Ala Ser
820 825 830
Leu Leu Glu Trp Glu Asp Ala Arg Asn Ile Ile Leu Arg Leu Gly Gln
835 840 845
Asn Pro Asp Glu Leu Leu Gly Ala Asp Pro Arg Arg His Ser Tyr Pro
850 855 860
Glu Ile Asn Asp Lys Leu Leu Val Ala Leu Arg Arg Ala Gln Ala Arg
865 870 875 880
Leu Ala Arg Leu Gln Asn Arg Ser Trp Arg Leu Arg Asp Leu Ala Glu
885 890 895
Ser Asp Lys Ala Leu Asp Glu Ile His Ala Glu Arg Ala Gly Glu Lys
900 905 910
Pro Ser Pro Leu Pro Pro Leu Ala Arg Asp Asp Ala Ile Lys Ser Thr
915 920 925
Asp Glu Ala Leu Leu Ser Gln Arg Asp Ile Ile Arg Arg Ser Phe Val
930 935 940
Gln Ile Ala Asn Leu Ile Leu Pro Leu Arg Gly Arg Arg Trp Glu Trp
945 950 955 960
Arg Pro His Val Glu Val Pro Asp Cys His Ile Leu Ala Gln Ser Asp
965 970 975
Pro Gly Thr Asp Asp Thr Lys Arg Leu Val Ala Gly Gln Arg Gly Ile
980 985 990
Ser His Glu Arg Ile Glu Gln Ile Glu Glu Leu Arg Arg Arg Cys Gln
995 1000 1005
Ser Leu Asn Arg Ala Leu Arg His Lys Pro Gly Glu Arg Pro Val
1010 1015 1020
Leu Gly Arg Pro Ala Lys Gly Glu Glu Ile Ala Asp Pro Cys Pro
1025 1030 1035
Ala Leu Leu Glu Lys Ile Asn Arg Leu Arg Asp Gln Arg Val Asp
1040 1045 1050
Gln Thr Ala His Ala Ile Leu Ala Ala Ala Leu Gly Val Arg Leu
1055 1060 1065
Arg Ala Pro Ser Lys Asp Arg Ala Glu Arg Arg His Arg Asp Ile
1070 1075 1080
His Gly Glu Tyr Glu Arg Phe Arg Ala Pro Ala Asp Phe Val Val
1085 1090 1095
Ile Glu Asn Leu Ser Arg Tyr Leu Ser Ser Gln Asp Arg Ala Arg
1100 1105 1110
Ser Glu Asn Thr Arg Leu Met Gln Trp Cys His Arg Gln Ile Val
1115 1120 1125
Gln Lys Leu Arg Gln Leu Cys Glu Thr Tyr Gly Ile Pro Val Leu
1130 1135 1140
Ala Val Pro Ala Ala Tyr Ser Ser Arg Phe Ser Ser Arg Asp Gly
1145 1150 1155
Ser Ala Gly Phe Arg Ala Val His Leu Thr Pro Asp His Arg His
1160 1165 1170
Arg Met Pro Trp Ser Arg Ile Leu Ala Arg Leu Lys Ala His Glu
1175 1180 1185
Glu Asp Gly Lys Arg Leu Glu Lys Thr Val Leu Asp Glu Ala Arg
1190 1195 1200
Ala Val Arg Gly Leu Phe Asp Arg Leu Asp Arg Phe Asn Ala Gly
1205 1210 1215
His Val Pro Gly Lys Pro Trp Arg Thr Leu Leu Ala Pro Leu Pro
1220 1225 1230
Gly Gly Pro Val Phe Val Pro Leu Gly Asp Ala Thr Pro Met Gln
1235 1240 1245
Ala Asp Leu Asn Ala Ala Ile Asn Ile Ala Leu Arg Gly Ile Ala
1250 1255 1260
Ala Pro Asp Arg His Asp Ile His His Arg Leu Arg Ala Glu Asn
1265 1270 1275
Lys Lys Arg Ile Leu Ser Leu Arg Leu Gly Thr Gln Arg Glu Lys
1280 1285 1290
Ala Arg Trp Pro Gly Gly Ala Pro Ala Val Thr Leu Ser Thr Pro
1295 1300 1305
Asn Asn Gly Ala Ser Pro Glu Asp Ser Asp Ala Leu Pro Glu Arg
1310 1315 1320
Val Ser Asn Leu Phe Val Asp Ile Ala Gly Val Ala Asn Phe Glu
1325 1330 1335
Arg Val Thr Ile Glu Gly Val Ser Gln Lys Phe Ala Thr Gly Arg
1340 1345 1350
Gly Leu Trp Ala Ser Val Lys Gln Arg Ala Trp Asn Arg Val Ala
1355 1360 1365
Arg Leu Asn Glu Thr Val Thr Asp Asn Asn Arg Asn Glu Glu Glu
1370 1375 1380
Asp Asp Ile Pro Met
1385
<210> 553
<211> 1132
<212> PRT
<213> Tuberibacillus calidus
<400> 553
Met Ala Thr Lys Ser Phe Ile Leu Lys Met Lys Thr Lys Asn Asn Pro
1 5 10 15
Gln Leu Arg Leu Ser Leu Trp Lys Thr His Glu Leu Phe Asn Phe Gly
20 25 30
Val Ala Tyr Tyr Met Asp Leu Leu Ser Leu Phe Arg Gln Lys Asp Leu
35 40 45
Tyr Met His Asn Asp Glu Asp Pro Asp His Pro Val Val Leu Lys Lys
50 55 60
Glu Glu Ile Gln Glu Arg Leu Trp Met Lys Val Arg Glu Thr Gln Gln
65 70 75 80
Lys Asn Gly Phe His Gly Glu Val Ser Lys Asp Glu Val Leu Glu Thr
85 90 95
Leu Arg Ala Leu Tyr Glu Glu Leu Val Pro Ser Ala Val Gly Lys Ser
100 105 110
Gly Glu Ala Asn Gln Ile Ser Asn Lys Tyr Leu Tyr Pro Leu Thr Asp
115 120 125
Pro Ala Ser Gln Ser Gly Lys Gly Thr Ala Asn Ser Gly Arg Lys Pro
130 135 140
Arg Trp Lys Lys Leu Lys Glu Ala Gly Asp Pro Ser Trp Lys Asp Ala
145 150 155 160
Tyr Glu Lys Trp Glu Lys Glu Arg Gln Glu Asp Pro Lys Leu Lys Ile
165 170 175
Leu Ala Ala Leu Gln Ser Phe Gly Leu Ile Pro Leu Phe Arg Pro Phe
180 185 190
Thr Glu Asn Asp His Lys Ala Val Ile Ser Val Lys Trp Met Pro Lys
195 200 205
Ser Lys Asn Gln Ser Val Arg Lys Phe Asp Lys Asp Met Phe Asn Gln
210 215 220
Ala Ile Glu Arg Phe Leu Ser Trp Glu Ser Trp Asn Glu Lys Val Ala
225 230 235 240
Glu Asp Tyr Glu Lys Thr Val Ser Ile Tyr Glu Ser Leu Gln Lys Glu
245 250 255
Leu Lys Gly Ile Ser Thr Lys Ala Phe Glu Ile Met Glu Arg Val Glu
260 265 270
Lys Ala Tyr Glu Ala His Leu Arg Glu Ile Thr Phe Ser Asn Ser Thr
275 280 285
Tyr Arg Ile Gly Asn Arg Ala Ile Arg Gly Trp Thr Glu Ile Val Lys
290 295 300
Lys Trp Met Lys Leu Asp Pro Ser Ala Pro Gln Gly Asn Tyr Leu Asp
305 310 315 320
Val Val Lys Asp Tyr Gln Arg Arg His Pro Arg Glu Ser Gly Asp Phe
325 330 335
Lys Leu Phe Glu Leu Leu Ser Arg Pro Glu Asn Gln Ala Ala Trp Arg
340 345 350
Glu Tyr Pro Glu Phe Leu Pro Leu Tyr Val Lys Tyr Arg His Ala Glu
355 360 365
Gln Arg Met Lys Thr Ala Lys Lys Gln Ala Thr Phe Thr Leu Cys Asp
370 375 380
Pro Ile Arg His Pro Leu Trp Val Arg Tyr Glu Glu Arg Ser Gly Thr
385 390 395 400
Asn Leu Asn Lys Tyr Arg Leu Ile Met Asn Glu Lys Glu Lys Val Val
405 410 415
Gln Phe Asp Arg Leu Ile Cys Leu Asn Ala Asp Gly His Tyr Glu Glu
420 425 430
Gln Glu Asp Val Thr Val Pro Leu Ala Pro Ser Gln Gln Phe Asp Asp
435 440 445
Gln Ile Lys Phe Ser Ser Glu Asp Thr Gly Lys Gly Lys His Asn Phe
450 455 460
Ser Tyr Tyr His Lys Gly Ile Asn Tyr Glu Leu Lys Gly Thr Leu Gly
465 470 475 480
Gly Ala Arg Ile Gln Phe Asp Arg Glu His Leu Leu Arg Arg Gln Gly
485 490 495
Val Lys Ala Gly Asn Val Gly Arg Ile Phe Leu Asn Val Thr Leu Asn
500 505 510
Ile Glu Pro Met Gln Pro Phe Ser Arg Ser Gly Asn Leu Gln Thr Ser
515 520 525
Val Gly Lys Ala Leu Lys Val Tyr Val Asp Gly Tyr Pro Lys Val Val
530 535 540
Asn Phe Lys Pro Lys Glu Leu Thr Glu His Ile Lys Glu Ser Glu Lys
545 550 555 560
Asn Thr Leu Thr Leu Gly Val Glu Ser Leu Pro Thr Gly Leu Arg Val
565 570 575
Met Ser Val Asp Leu Gly Gln Arg Gln Ala Ala Ala Ile Ser Ile Phe
580 585 590
Glu Val Val Ser Glu Lys Pro Asp Asp Asn Lys Leu Phe Tyr Pro Val
595 600 605
Lys Asp Thr Asp Leu Phe Ala Val His Arg Thr Ser Phe Asn Ile Lys
610 615 620
Leu Pro Gly Glu Lys Arg Thr Glu Arg Arg Met Leu Glu Gln Gln Lys
625 630 635 640
Arg Asp Gln Ala Ile Arg Asp Leu Ser Arg Lys Leu Lys Phe Leu Lys
645 650 655
Asn Val Leu Asn Met Gln Lys Leu Glu Lys Thr Asp Glu Arg Glu Lys
660 665 670
Arg Val Asn Arg Trp Ile Lys Asp Arg Glu Arg Glu Glu Glu Asn Pro
675 680 685
Val Tyr Val Gln Glu Phe Glu Met Ile Ser Lys Val Leu Tyr Ser Pro
690 695 700
His Ser Val Trp Val Asp Gln Leu Lys Ser Ile His Arg Lys Leu Glu
705 710 715 720
Glu Gln Leu Gly Lys Glu Ile Ser Lys Trp Arg Gln Ser Ile Ser Gln
725 730 735
Gly Arg Gln Gly Val Tyr Gly Ile Ser Leu Lys Asn Ile Glu Asp Ile
740 745 750
Glu Lys Thr Arg Arg Leu Leu Phe Arg Trp Ser Met Arg Pro Glu Asn
755 760 765
Pro Gly Glu Val Lys Gln Leu Gln Pro Gly Glu Arg Phe Ala Ile Asp
770 775 780
Gln Gln Asn His Leu Asn His Leu Lys Asp Asp Arg Ile Lys Lys Leu
785 790 795 800
Ala Asn Gln Ile Val Met Thr Ala Leu Gly Tyr Arg Tyr Asp Gly Lys
805 810 815
Arg Lys Lys Trp Ile Ala Lys His Pro Ala Cys Gln Leu Val Leu Phe
820 825 830
Glu Asp Leu Ser Arg Tyr Ala Phe Tyr Asp Glu Arg Ser Arg Leu Glu
835 840 845
Asn Arg Asn Leu Met Arg Trp Ser Arg Arg Glu Ile Pro Lys Gln Val
850 855 860
Ala Gln Ile Gly Gly Leu Tyr Gly Leu Leu Val Gly Glu Val Gly Ala
865 870 875 880
Gln Tyr Ser Ser Arg Phe His Ala Lys Ser Gly Ala Pro Gly Ile Arg
885 890 895
Cys Arg Val Val Lys Glu His Glu Leu Tyr Ile Thr Glu Gly Gly Gln
900 905 910
Lys Val Arg Asn Gln Lys Phe Leu Asp Ser Leu Val Glu Asn Asn Ile
915 920 925
Ile Glu Pro Asp Asp Ala Arg Arg Leu Glu Pro Gly Asp Leu Ile Arg
930 935 940
Asp Gln Gly Gly Asp Lys Phe Ala Thr Leu Asp Glu Arg Gly Glu Leu
945 950 955 960
Val Ile Thr His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln Lys Arg
965 970 975
Phe Trp Thr Arg Thr His Gly Leu Tyr Arg Ile Arg Cys Glu Ser Arg
980 985 990
Glu Ile Lys Asp Ala Val Val Leu Val Pro Ser Asp Lys Asp Gln Lys
995 1000 1005
Glu Lys Met Glu Asn Leu Phe Gly Ile Gly Tyr Leu Gln Pro Phe
1010 1015 1020
Lys Gln Glu Asn Asp Val Tyr Lys Trp Val Lys Gly Glu Lys Ile
1025 1030 1035
Lys Gly Lys Lys Thr Ser Ser Gln Ser Asp Asp Lys Glu Leu Val
1040 1045 1050
Ser Glu Ile Leu Gln Glu Ala Ser Val Met Ala Asp Glu Leu Lys
1055 1060 1065
Gly Asn Arg Lys Thr Leu Phe Arg Asp Pro Ser Gly Tyr Val Phe
1070 1075 1080
Pro Lys Asp Arg Trp Tyr Thr Gly Gly Arg Tyr Phe Gly Thr Leu
1085 1090 1095
Glu His Leu Leu Lys Arg Lys Leu Ala Glu Arg Arg Leu Phe Asp
1100 1105 1110
Gly Gly Ser Ser Arg Arg Gly Leu Phe Asn Gly Thr Asp Ser Asn
1115 1120 1125
Thr Asn Val Glu
1130
<210> 554
<211> 1108
<212> PRT
<213> Bacillus thermoamylovorans
<400> 554
Met Ala Thr Arg Ser Phe Ile Leu Lys Ile Glu Pro Asn Glu Glu Val
1 5 10 15
Lys Lys Gly Leu Trp Lys Thr His Glu Val Leu Asn His Gly Ile Ala
20 25 30
Tyr Tyr Met Asn Ile Leu Lys Leu Ile Arg Gln Glu Ala Ile Tyr Glu
35 40 45
His His Glu Gln Asp Pro Lys Asn Pro Lys Lys Val Ser Lys Ala Glu
50 55 60
Ile Gln Ala Glu Leu Trp Asp Phe Val Leu Lys Met Gln Lys Cys Asn
65 70 75 80
Ser Phe Thr His Glu Val Asp Lys Asp Val Val Phe Asn Ile Leu Arg
85 90 95
Glu Leu Tyr Glu Glu Leu Val Pro Ser Ser Val Glu Lys Lys Gly Glu
100 105 110
Ala Asn Gln Leu Ser Asn Lys Phe Leu Tyr Pro Leu Val Asp Pro Asn
115 120 125
Ser Gln Ser Gly Lys Gly Thr Ala Ser Ser Gly Arg Lys Pro Arg Trp
130 135 140
Tyr Asn Leu Lys Ile Ala Gly Asp Pro Ser Trp Glu Glu Glu Lys Lys
145 150 155 160
Lys Trp Glu Glu Asp Lys Lys Lys Asp Pro Leu Ala Lys Ile Leu Gly
165 170 175
Lys Leu Ala Glu Tyr Gly Leu Ile Pro Leu Phe Ile Pro Phe Thr Asp
180 185 190
Ser Asn Glu Pro Ile Val Lys Glu Ile Lys Trp Met Glu Lys Ser Arg
195 200 205
Asn Gln Ser Val Arg Arg Leu Asp Lys Asp Met Phe Ile Gln Ala Leu
210 215 220
Glu Arg Phe Leu Ser Trp Glu Ser Trp Asn Leu Lys Val Lys Glu Glu
225 230 235 240
Tyr Glu Lys Val Glu Lys Glu His Lys Thr Leu Glu Glu Arg Ile Lys
245 250 255
Glu Asp Ile Gln Ala Phe Lys Ser Leu Glu Gln Tyr Glu Lys Glu Arg
260 265 270
Gln Glu Gln Leu Leu Arg Asp Thr Leu Asn Thr Asn Glu Tyr Arg Leu
275 280 285
Ser Lys Arg Gly Leu Arg Gly Trp Arg Glu Ile Ile Gln Lys Trp Leu
290 295 300
Lys Met Asp Glu Asn Glu Pro Ser Glu Lys Tyr Leu Glu Val Phe Lys
305 310 315 320
Asp Tyr Gln Arg Lys His Pro Arg Glu Ala Gly Asp Tyr Ser Val Tyr
325 330 335
Glu Phe Leu Ser Lys Lys Glu Asn His Phe Ile Trp Arg Asn His Pro
340 345 350
Glu Tyr Pro Tyr Leu Tyr Ala Thr Phe Cys Glu Ile Asp Lys Lys Lys
355 360 365
Lys Asp Ala Lys Gln Gln Ala Thr Phe Thr Leu Ala Asp Pro Ile Asn
370 375 380
His Pro Leu Trp Val Arg Phe Glu Glu Arg Ser Gly Ser Asn Leu Asn
385 390 395 400
Lys Tyr Arg Ile Leu Thr Glu Gln Leu His Thr Glu Lys Leu Lys Lys
405 410 415
Lys Leu Thr Val Gln Leu Asp Arg Leu Ile Tyr Pro Thr Glu Ser Gly
420 425 430
Gly Trp Glu Glu Lys Gly Lys Val Asp Ile Val Leu Leu Pro Ser Arg
435 440 445
Gln Phe Tyr Asn Gln Ile Phe Leu Asp Ile Glu Glu Lys Gly Lys His
450 455 460
Ala Phe Thr Tyr Lys Asp Glu Ser Ile Lys Phe Pro Leu Lys Gly Thr
465 470 475 480
Leu Gly Gly Ala Arg Val Gln Phe Asp Arg Asp His Leu Arg Arg Tyr
485 490 495
Pro His Lys Val Glu Ser Gly Asn Val Gly Arg Ile Tyr Phe Asn Met
500 505 510
Thr Val Asn Ile Glu Pro Thr Glu Ser Pro Val Ser Lys Ser Leu Lys
515 520 525
Ile His Arg Asp Asp Phe Pro Lys Phe Val Asn Phe Lys Pro Lys Glu
530 535 540
Leu Thr Glu Trp Ile Lys Asp Ser Lys Gly Lys Lys Leu Lys Ser Gly
545 550 555 560
Ile Glu Ser Leu Glu Ile Gly Leu Arg Val Met Ser Ile Asp Leu Gly
565 570 575
Gln Arg Gln Ala Ala Ala Ala Ser Ile Phe Glu Val Val Asp Gln Lys
580 585 590
Pro Asp Ile Glu Gly Lys Leu Phe Phe Pro Ile Lys Gly Thr Glu Leu
595 600 605
Tyr Ala Val His Arg Ala Ser Phe Asn Ile Lys Leu Pro Gly Glu Thr
610 615 620
Leu Val Lys Ser Arg Glu Val Leu Arg Lys Ala Arg Glu Asp Asn Leu
625 630 635 640
Lys Leu Met Asn Gln Lys Leu Asn Phe Leu Arg Asn Val Leu His Phe
645 650 655
Gln Gln Phe Glu Asp Ile Thr Glu Arg Glu Lys Arg Val Thr Lys Trp
660 665 670
Ile Ser Arg Gln Glu Asn Ser Asp Val Pro Leu Val Tyr Gln Asp Glu
675 680 685
Leu Ile Gln Ile Arg Glu Leu Met Tyr Lys Pro Tyr Lys Asp Trp Val
690 695 700
Ala Phe Leu Lys Gln Leu His Lys Arg Leu Glu Val Glu Ile Gly Lys
705 710 715 720
Glu Val Lys His Trp Arg Lys Ser Leu Ser Asp Gly Arg Lys Gly Leu
725 730 735
Tyr Gly Ile Ser Leu Lys Asn Ile Asp Glu Ile Asp Arg Thr Arg Lys
740 745 750
Phe Leu Leu Arg Trp Ser Leu Arg Pro Thr Glu Pro Gly Glu Val Arg
755 760 765
Arg Leu Glu Pro Gly Gln Arg Phe Ala Ile Asp Gln Leu Asn His Leu
770 775 780
Asn Ala Leu Lys Glu Asp Arg Leu Lys Lys Met Ala Asn Thr Ile Ile
785 790 795 800
Met His Ala Leu Gly Tyr Cys Tyr Asp Val Arg Lys Lys Lys Trp Gln
805 810 815
Ala Lys Asn Pro Ala Cys Gln Ile Ile Leu Phe Glu Asp Leu Ser Asn
820 825 830
Tyr Asn Pro Tyr Glu Glu Arg Ser Arg Phe Glu Asn Ser Lys Leu Met
835 840 845
Lys Trp Ser Arg Arg Glu Ile Pro Arg Gln Val Ala Leu Gln Gly Glu
850 855 860
Ile Tyr Gly Leu Gln Val Gly Glu Val Gly Ala Gln Phe Ser Ser Arg
865 870 875 880
Phe His Ala Lys Thr Gly Ser Pro Gly Ile Arg Cys Ser Val Val Thr
885 890 895
Lys Glu Lys Leu Gln Asp Asn Arg Phe Phe Lys Asn Leu Gln Arg Glu
900 905 910
Gly Arg Leu Thr Leu Asp Lys Ile Ala Val Leu Lys Glu Gly Asp Leu
915 920 925
Tyr Pro Asp Lys Gly Gly Glu Lys Phe Ile Ser Leu Ser Lys Asp Arg
930 935 940
Lys Leu Val Thr Thr His Ala Asp Ile Asn Ala Ala Gln Asn Leu Gln
945 950 955 960
Lys Arg Phe Trp Thr Arg Thr His Gly Phe Tyr Lys Val Tyr Cys Lys
965 970 975
Ala Tyr Gln Val Asp Gly Gln Thr Val Tyr Ile Pro Glu Ser Lys Asp
980 985 990
Gln Lys Gln Lys Ile Ile Glu Glu Phe Gly Glu Gly Tyr Phe Ile Leu
995 1000 1005
Lys Asp Gly Val Tyr Glu Trp Gly Asn Ala Gly Lys Leu Lys Ile
1010 1015 1020
Lys Lys Gly Ser Ser Lys Gln Ser Ser Ser Glu Leu Val Asp Ser
1025 1030 1035
Asp Ile Leu Lys Asp Ser Phe Asp Leu Ala Ser Glu Leu Lys Gly
1040 1045 1050
Glu Lys Leu Met Leu Tyr Arg Asp Pro Ser Gly Asn Val Phe Pro
1055 1060 1065
Ser Asp Lys Trp Met Ala Ala Gly Val Phe Phe Gly Lys Leu Glu
1070 1075 1080
Arg Ile Leu Ile Ser Lys Leu Thr Asn Gln Tyr Ser Ile Ser Thr
1085 1090 1095
Ile Glu Asp Asp Ser Ser Lys Gln Ser Met
1100 1105
<210> 555
<211> 864
<212> PRT
<213> Brevibacillus sp.
<400> 555
Met Pro Lys Ile Leu Arg Gly His Lys Trp Ile Ser Leu Leu Glu Gln
1 5 10 15
Tyr Glu Glu Asn Arg Glu Arg Glu Leu Arg Glu Asn Met Thr Ala Ala
20 25 30
Asn Asp Lys Tyr Arg Ile Thr Lys Arg Gln Met Lys Gly Trp Asn Glu
35 40 45
Leu Tyr Glu Leu Trp Ser Thr Phe Pro Ala Ser Ala Ser His Glu Gln
50 55 60
Tyr Lys Glu Ala Leu Lys Arg Val Gln Gln Arg Leu Arg Gly Arg Phe
65 70 75 80
Gly Asp Ala His Phe Phe Gln Tyr Leu Met Glu Glu Lys Asn Arg Leu
85 90 95
Ile Trp Lys Gly Asn Pro Gln Arg Ile His Tyr Phe Val Ala Arg Asn
100 105 110
Glu Leu Thr Lys Arg Leu Glu Glu Ala Lys Gln Ser Ala Thr Met Thr
115 120 125
Leu Pro Asn Ala Arg Lys His Pro Leu Trp Val Arg Phe Asp Ala Arg
130 135 140
Gly Gly Asn Leu Gln Asp Tyr Tyr Leu Thr Ala Glu Ala Asp Lys Pro
145 150 155 160
Arg Ser Arg Arg Phe Val Thr Phe Ser Gln Leu Ile Trp Pro Ser Glu
165 170 175
Ser Gly Trp Met Glu Lys Lys Asp Val Glu Val Glu Leu Ala Leu Ser
180 185 190
Arg Gln Phe Tyr Gln Gln Val Lys Leu Leu Lys Asn Asp Lys Gly Lys
195 200 205
Gln Lys Ile Glu Phe Lys Asp Lys Gly Ser Gly Ser Thr Phe Asn Gly
210 215 220
His Leu Gly Gly Ala Lys Leu Gln Leu Glu Arg Gly Asp Leu Glu Lys
225 230 235 240
Glu Glu Lys Asn Phe Glu Asp Gly Glu Ile Gly Ser Val Tyr Leu Asn
245 250 255
Val Val Ile Asp Phe Glu Pro Leu Gln Glu Val Lys Asn Gly Arg Val
260 265 270
Gln Ala Pro Tyr Gly Gln Val Leu Gln Leu Ile Arg Arg Pro Asn Glu
275 280 285
Phe Pro Lys Val Thr Thr Tyr Lys Ser Glu Gln Leu Val Glu Trp Ile
290 295 300
Lys Ala Ser Pro Gln His Ser Ala Gly Val Glu Ser Leu Ala Ser Gly
305 310 315 320
Phe Arg Val Met Ser Ile Asp Leu Gly Leu Arg Ala Ala Ala Ala Thr
325 330 335
Ser Ile Phe Ser Val Glu Glu Ser Ser Asp Lys Asn Ala Ala Asp Phe
340 345 350
Ser Tyr Trp Ile Glu Gly Thr Pro Leu Val Ala Val His His Arg Ser
355 360 365
Tyr Met Leu Arg Leu Pro Gly Glu Gln Val Glu Lys Gln Val Met Glu
370 375 380
Lys Arg Asp Glu Arg Phe Gln Leu His Gln Arg Val Lys Phe Gln Ile
385 390 395 400
Arg Val Leu Ala Gln Ile Met Arg Met Ala Asn Lys Gln Tyr Gly Asp
405 410 415
Arg Trp Asp Glu Leu Asp Ser Leu Lys Gln Ala Val Glu Gln Lys Lys
420 425 430
Ser Pro Leu Asp Gln Thr Asp Arg Thr Phe Trp Glu Gly Ile Val Cys
435 440 445
Asp Leu Thr Lys Val Leu Pro Arg Asn Glu Ala Asp Trp Glu Gln Ala
450 455 460
Val Val Gln Ile His Arg Lys Ala Glu Glu Tyr Val Gly Lys Ala Val
465 470 475 480
Gln Ala Trp Arg Lys Arg Phe Ala Ala Asp Glu Arg Lys Gly Ile Ala
485 490 495
Gly Leu Ser Met Trp Asn Ile Glu Glu Leu Glu Gly Leu Arg Lys Leu
500 505 510
Leu Ile Ser Trp Ser Arg Arg Ser Arg Asn Pro Gln Glu Val Asn Arg
515 520 525
Phe Glu Arg Gly His Thr Ser His Gln Arg Leu Leu Thr His Ile Gln
530 535 540
Asn Val Lys Glu Asp Arg Leu Lys Gln Leu Ser His Ala Ile Val Met
545 550 555 560
Thr Ala Leu Gly Tyr Val Tyr Asp Glu Arg Lys Gln Glu Trp Cys Ala
565 570 575
Glu Tyr Pro Ala Cys Gln Val Ile Leu Phe Glu Asn Leu Ser Gln Tyr
580 585 590
Arg Ser Asn Leu Asp Arg Ser Thr Lys Glu Asn Ser Thr Leu Met Lys
595 600 605
Trp Ala His Arg Ser Ile Pro Lys Tyr Val His Met Gln Ala Glu Pro
610 615 620
Tyr Gly Ile Gln Ile Gly Asp Val Arg Ala Glu Tyr Ser Ser Arg Phe
625 630 635 640
Tyr Ala Lys Thr Gly Thr Pro Gly Ile Arg Cys Lys Lys Val Arg Gly
645 650 655
Gln Asp Leu Gln Gly Arg Arg Phe Glu Asn Leu Gln Lys Arg Leu Val
660 665 670
Asn Glu Gln Phe Leu Thr Glu Glu Gln Val Lys Gln Leu Arg Pro Gly
675 680 685
Asp Ile Val Pro Asp Asp Ser Gly Glu Leu Phe Met Thr Leu Thr Asp
690 695 700
Gly Ser Gly Ser Lys Glu Val Val Phe Leu Gln Ala Asp Ile Asn Ala
705 710 715 720
Ala His Asn Leu Gln Lys Arg Phe Trp Gln Arg Tyr Asn Glu Leu Phe
725 730 735
Lys Val Ser Cys Arg Val Ile Val Arg Asp Glu Glu Glu Tyr Leu Val
740 745 750
Pro Lys Thr Lys Ser Val Gln Ala Lys Leu Gly Lys Gly Leu Phe Val
755 760 765
Lys Lys Ser Asp Thr Ala Trp Lys Asp Val Tyr Val Trp Asp Ser Gln
770 775 780
Ala Lys Leu Lys Gly Lys Thr Thr Phe Thr Glu Glu Ser Glu Ser Pro
785 790 795 800
Glu Gln Leu Glu Asp Phe Gln Glu Ile Ile Glu Glu Ala Glu Glu Ala
805 810 815
Lys Gly Thr Tyr Arg Thr Leu Phe Arg Asp Pro Ser Gly Val Phe Phe
820 825 830
Pro Glu Ser Val Trp Tyr Pro Gln Lys Asp Phe Trp Gly Glu Val Lys
835 840 845
Arg Lys Leu Tyr Gly Lys Leu Arg Glu Arg Phe Leu Thr Lys Ala Arg
850 855 860
<210> 556
<211> 1108
<212> PRT
<213> Bacillus sp.
<400> 556
Met Ala Ile Arg Ser Ile Lys Leu Lys Leu Lys Thr His Thr Gly Pro
1 5 10 15
Glu Ala Gln Asn Leu Arg Lys Gly Ile Trp Arg Thr His Arg Leu Leu
20 25 30
Asn Glu Gly Val Ala Tyr Tyr Met Lys Met Leu Leu Leu Phe Arg Gln
35 40 45
Glu Ser Thr Gly Glu Arg Pro Lys Glu Glu Leu Gln Glu Glu Leu Ile
50 55 60
Cys His Ile Arg Glu Gln Gln Gln Arg Asn Gln Ala Asp Lys Asn Thr
65 70 75 80
Gln Ala Leu Pro Leu Asp Lys Ala Leu Glu Ala Leu Arg Gln Leu Tyr
85 90 95
Glu Leu Leu Val Pro Ser Ser Val Gly Gln Ser Gly Asp Ala Gln Ile
100 105 110
Ile Ser Arg Lys Phe Leu Ser Pro Leu Val Asp Pro Asn Ser Glu Gly
115 120 125
Gly Lys Gly Thr Ser Lys Ala Gly Ala Lys Pro Thr Trp Gln Lys Lys
130 135 140
Lys Glu Ala Asn Asp Pro Thr Trp Glu Gln Asp Tyr Glu Lys Trp Lys
145 150 155 160
Lys Arg Arg Glu Glu Asp Pro Thr Ala Ser Val Ile Thr Thr Leu Glu
165 170 175
Glu Tyr Gly Ile Arg Pro Ile Phe Pro Leu Tyr Thr Asn Thr Val Thr
180 185 190
Asp Ile Ala Trp Leu Pro Leu Gln Ser Asn Gln Phe Val Arg Thr Trp
195 200 205
Asp Arg Asp Met Leu Gln Gln Ala Ile Glu Arg Leu Leu Ser Trp Glu
210 215 220
Ser Trp Asn Lys Arg Val Gln Glu Glu Tyr Ala Lys Leu Lys Glu Lys
225 230 235 240
Met Ala Gln Leu Asn Glu Gln Leu Glu Gly Gly Gln Glu Trp Ile Ser
245 250 255
Leu Leu Glu Gln Tyr Glu Glu Asn Arg Glu Arg Glu Leu Arg Glu Asn
260 265 270
Met Thr Ala Ala Asn Asp Lys Tyr Arg Ile Thr Lys Arg Gln Met Lys
275 280 285
Gly Trp Asn Glu Leu Tyr Glu Leu Trp Ser Thr Phe Pro Ala Ser Ala
290 295 300
Ser His Glu Gln Tyr Lys Glu Ala Leu Lys Arg Val Gln Gln Arg Leu
305 310 315 320
Arg Gly Arg Phe Gly Asp Ala His Phe Phe Gln Tyr Leu Met Glu Glu
325 330 335
Lys Asn Arg Leu Ile Trp Lys Gly Asn Pro Gln Arg Ile His Tyr Phe
340 345 350
Val Ala Arg Asn Glu Leu Thr Lys Arg Leu Glu Glu Ala Lys Gln Ser
355 360 365
Ala Thr Met Thr Leu Pro Asn Ala Arg Lys His Pro Leu Trp Val Arg
370 375 380
Phe Asp Ala Arg Gly Gly Asn Leu Gln Asp Tyr Tyr Leu Thr Ala Glu
385 390 395 400
Ala Asp Lys Pro Arg Ser Arg Arg Phe Val Thr Phe Ser Gln Leu Ile
405 410 415
Trp Pro Ser Glu Ser Gly Trp Met Glu Lys Lys Asp Val Glu Val Glu
420 425 430
Leu Ala Leu Ser Arg Gln Phe Tyr Gln Gln Val Lys Leu Leu Lys Asn
435 440 445
Asp Lys Gly Lys Gln Lys Ile Glu Phe Lys Asp Lys Gly Ser Gly Ser
450 455 460
Thr Phe Asn Gly His Leu Gly Gly Ala Lys Leu Gln Leu Glu Arg Gly
465 470 475 480
Asp Leu Glu Lys Glu Glu Lys Asn Phe Glu Asp Gly Glu Ile Gly Ser
485 490 495
Val Tyr Leu Asn Val Val Ile Asp Phe Glu Pro Leu Gln Glu Val Lys
500 505 510
Asn Gly Arg Val Gln Ala Pro Tyr Gly Gln Val Leu Gln Leu Ile Arg
515 520 525
Arg Pro Asn Glu Phe Pro Lys Val Thr Thr Tyr Lys Ser Glu Gln Leu
530 535 540
Val Glu Trp Ile Lys Ala Ser Pro Gln His Ser Ala Gly Val Glu Ser
545 550 555 560
Leu Ala Ser Gly Phe Arg Val Met Ser Ile Asp Leu Gly Leu Arg Ala
565 570 575
Ala Ala Ala Thr Ser Ile Phe Ser Val Glu Glu Ser Ser Asp Lys Asn
580 585 590
Ala Ala Asp Phe Ser Tyr Trp Ile Glu Gly Thr Pro Leu Val Ala Val
595 600 605
His Gln Arg Ser Tyr Met Leu Arg Leu Pro Gly Glu Gln Val Glu Lys
610 615 620
Gln Val Met Glu Lys Arg Asp Glu Arg Phe Gln Leu His Gln Arg Val
625 630 635 640
Lys Phe Gln Ile Arg Val Leu Ala Gln Ile Met Arg Met Ala Asn Lys
645 650 655
Gln Tyr Gly Asp Arg Trp Asp Glu Leu Asp Ser Leu Lys Gln Ala Val
660 665 670
Glu Gln Lys Lys Ser Pro Leu Asp Gln Thr Asp Arg Thr Phe Trp Glu
675 680 685
Gly Ile Val Cys Asp Leu Thr Lys Val Leu Pro Arg Asn Glu Ala Asp
690 695 700
Trp Glu Gln Ala Val Val Gln Ile His Arg Lys Ala Glu Glu Tyr Val
705 710 715 720
Gly Lys Ala Val Gln Ala Trp Arg Lys Arg Phe Ala Ala Asp Glu Arg
725 730 735
Lys Gly Ile Ala Gly Leu Ser Met Trp Asn Ile Glu Glu Leu Glu Gly
740 745 750
Leu Arg Lys Leu Leu Ile Ser Trp Ser Arg Arg Thr Arg Asn Pro Gln
755 760 765
Glu Val Asn Arg Phe Glu Arg Gly His Thr Ser His Gln Arg Leu Leu
770 775 780
Thr His Ile Gln Asn Val Lys Glu Asp Arg Leu Lys Gln Leu Ser His
785 790 795 800
Ala Ile Val Met Thr Ala Leu Gly Tyr Val Tyr Asp Glu Arg Lys Gln
805 810 815
Glu Trp Cys Ala Glu Tyr Pro Ala Cys Gln Val Ile Leu Phe Glu Asn
820 825 830
Leu Ser Gln Tyr Arg Ser Asn Leu Asp Arg Ser Thr Lys Glu Asn Ser
835 840 845
Thr Leu Met Lys Trp Ala His Arg Ser Ile Pro Lys Tyr Val His Met
850 855 860
Gln Ala Glu Pro Tyr Gly Ile Gln Ile Gly Asp Val Arg Ala Glu Tyr
865 870 875 880
Ser Ser Arg Phe Tyr Ala Lys Thr Gly Thr Pro Gly Ile Arg Cys Lys
885 890 895
Lys Val Arg Gly Gln Asp Leu Gln Gly Arg Arg Phe Glu Asn Leu Gln
900 905 910
Lys Arg Leu Val Asn Glu Gln Phe Leu Thr Glu Glu Gln Val Lys Gln
915 920 925
Leu Arg Pro Gly Asp Ile Val Pro Asp Asp Ser Gly Glu Leu Phe Met
930 935 940
Thr Leu Thr Asp Gly Ser Gly Ser Lys Glu Val Val Phe Leu Gln Ala
945 950 955 960
Asp Ile Asn Ala Ala His Asn Leu Gln Lys Arg Phe Trp Gln Arg Tyr
965 970 975
Asn Glu Leu Phe Lys Val Ser Cys Arg Val Ile Val Arg Asp Glu Glu
980 985 990
Glu Tyr Leu Val Pro Lys Thr Lys Ser Val Gln Ala Lys Leu Gly Lys
995 1000 1005
Gly Leu Phe Val Lys Lys Ser Asp Thr Ala Trp Lys Asp Val Tyr
1010 1015 1020
Val Trp Asp Ser Gln Ala Lys Leu Lys Gly Lys Thr Thr Phe Thr
1025 1030 1035
Glu Glu Ser Glu Ser Pro Glu Gln Leu Glu Asp Phe Gln Glu Ile
1040 1045 1050
Ile Glu Glu Ala Glu Glu Ala Lys Gly Thr Tyr Arg Thr Leu Phe
1055 1060 1065
Arg Asp Pro Ser Gly Val Phe Phe Pro Glu Ser Val Trp Tyr Pro
1070 1075 1080
Gln Lys Asp Phe Trp Gly Glu Val Lys Arg Lys Leu Tyr Gly Lys
1085 1090 1095
Leu Arg Glu Arg Phe Leu Thr Lys Ala Arg
1100 1105
<210> 557
<211> 1489
<212> PRT
<213> Desulfatirhabdium butyrativorans
<400> 557
Met Pro Leu Ser Asn Asn Pro Pro Val Thr Gln Arg Ala Tyr Thr Leu
1 5 10 15
Arg Leu Arg Gly Ala Asp Pro Ser Asp Leu Ser Trp Arg Glu Ala Leu
20 25 30
Trp His Thr His Glu Ala Val Asn Lys Gly Ala Lys Val Phe Gly Asp
35 40 45
Trp Leu Leu Thr Leu Arg Gly Gly Leu Asp His Thr Leu Ala Asp Thr
50 55 60
Lys Val Lys Gly Gly Lys Gly Lys Pro Asp Arg Asp Pro Thr Pro Glu
65 70 75 80
Glu Arg Lys Ala Arg Arg Ile Leu Leu Ala Leu Ser Trp Leu Ser Val
85 90 95
Glu Ser Lys Leu Gly Ala Pro Ser Ser Tyr Ile Val Ala Ser Gly Asp
100 105 110
Glu Pro Ala Lys Asp Arg Asn Asp Asn Val Val Ser Ala Leu Glu Glu
115 120 125
Ile Leu Gln Ser Arg Lys Val Ala Lys Ser Glu Ile Asp Asp Trp Lys
130 135 140
Arg Asp Cys Ser Ala Ser Leu Ser Ala Ala Ile Arg Asp Asp Ala Val
145 150 155 160
Trp Val Asn Arg Ser Lys Val Phe Asp Glu Ala Val Lys Ser Val Gly
165 170 175
Ser Ser Leu Thr Arg Glu Glu Ala Trp Asp Met Leu Glu Arg Phe Phe
180 185 190
Gly Ser Arg Asp Ala Tyr Leu Thr Pro Met Lys Asp Pro Glu Asp Lys
195 200 205
Ser Ser Glu Thr Glu Gln Glu Asp Lys Ala Lys Asp Leu Val Gln Lys
210 215 220
Ala Gly Gln Trp Leu Ser Ser Arg Tyr Gly Thr Ser Glu Gly Ala Asp
225 230 235 240
Phe Cys Arg Met Ser Asp Ile Tyr Gly Lys Ile Ala Ala Trp Ala Asp
245 250 255
Asn Ala Ser Gln Gly Gly Ser Ser Thr Val Asp Asp Leu Val Ser Glu
260 265 270
Leu Arg Gln His Phe Asp Thr Lys Glu Ser Lys Ala Thr Asn Gly Leu
275 280 285
Asp Trp Ile Ile Gly Leu Ser Ser Tyr Thr Gly His Thr Pro Asn Pro
290 295 300
Val His Glu Leu Leu Arg Gln Asn Thr Ser Leu Asn Lys Ser His Leu
305 310 315 320
Asp Asp Leu Lys Lys Lys Ala Asn Thr Arg Ala Glu Ser Cys Lys Ser
325 330 335
Lys Ile Gly Ser Lys Gly Gln Arg Pro Tyr Ser Asp Ala Ile Leu Asn
340 345 350
Asp Val Glu Ser Val Cys Gly Phe Thr Tyr Arg Val Asp Lys Asp Gly
355 360 365
Gln Pro Val Ser Val Ala Asp Tyr Ser Lys Tyr Asp Val Asp Tyr Lys
370 375 380
Trp Gly Thr Ala Arg His Tyr Ile Phe Ala Val Met Leu Asp His Ala
385 390 395 400
Ala Arg Arg Ile Ser Leu Ala His Lys Trp Ile Lys Arg Ala Glu Ala
405 410 415
Glu Arg His Lys Phe Glu Glu Asp Ala Lys Arg Ile Ala Asn Val Pro
420 425 430
Ala Arg Ala Arg Glu Trp Leu Asp Ser Phe Cys Lys Glu Arg Ser Val
435 440 445
Thr Ser Gly Ala Val Glu Pro Tyr Arg Ile Arg Arg Arg Ala Val Asp
450 455 460
Gly Trp Lys Glu Val Val Ala Ala Trp Ser Lys Ser Asp Cys Lys Ser
465 470 475 480
Thr Glu Asp Arg Ile Ala Ala Ala Arg Ala Leu Gln Asp Asp Ser Glu
485 490 495
Ile Asp Lys Phe Gly Asp Ile Gln Leu Phe Glu Ala Leu Ala Glu Asp
500 505 510
Asp Ala Leu Cys Val Trp His Lys Asp Gly Glu Ala Thr Asn Glu Pro
515 520 525
Asp Phe Gln Pro Leu Ile Asp Tyr Ser Leu Ala Ile Glu Ala Glu Phe
530 535 540
Lys Lys Arg Gln Phe Lys Val Pro Ala Tyr Arg His Pro Asp Glu Leu
545 550 555 560
Leu His Pro Val Phe Cys Asp Phe Gly Lys Ser Arg Trp Lys Ile Asn
565 570 575
Tyr Asp Val His Lys Asn Val Gln Ala Pro Phe Tyr Arg Gly Leu Cys
580 585 590
Leu Thr Leu Trp Thr Gly Ser Glu Ile Lys Pro Val Pro Leu Cys Trp
595 600 605
Gln Ser Lys Arg Leu Thr Arg Asp Leu Ala Leu Gly Asn Asn His Arg
610 615 620
Asn Asp Ala Ala Ser Ala Val Thr Arg Ala Asp Arg Leu Gly Arg Ala
625 630 635 640
Ala Ser Asn Val Thr Lys Ser Asp Met Val Asn Ile Thr Gly Leu Phe
645 650 655
Glu Gln Ala Asp Trp Asn Gly Arg Leu Gln Ala Pro Arg Gln Gln Leu
660 665 670
Glu Ala Ile Ala Val Val Arg Asp Asn Pro Arg Leu Ser Glu Gln Glu
675 680 685
Arg Asn Leu Arg Met Cys Gly Met Ile Glu His Ile Arg Trp Leu Val
690 695 700
Thr Phe Ser Val Lys Leu Gln Pro Gln Gly Pro Trp Cys Ala Tyr Ala
705 710 715 720
Glu Gln His Gly Leu Asn Thr Asn Pro Gln Tyr Trp Pro His Ala Asp
725 730 735
Thr Asn Arg Asp Arg Lys Val His Ala Arg Leu Ile Leu Pro Arg Leu
740 745 750
Pro Gly Leu Arg Val Leu Ser Val Asp Leu Gly His Arg Tyr Ala Ala
755 760 765
Ala Cys Ala Val Trp Glu Ala Val Asn Thr Glu Thr Val Lys Glu Ala
770 775 780
Cys Gln Asn Val Gly Arg Asp Met Pro Lys Glu His Asp Leu Tyr Leu
785 790 795 800
His Ile Lys Val Lys Lys Gln Gly Ile Gly Lys Gln Thr Glu Val Asp
805 810 815
Lys Thr Thr Ile Tyr Arg Arg Ile Gly Ala Asp Thr Leu Pro Asp Gly
820 825 830
Arg Pro His Pro Ala Pro Trp Ala Arg Leu Asp Arg Gln Phe Leu Ile
835 840 845
Lys Leu Gln Gly Glu Glu Lys Asp Ala Arg Glu Ala Ser Asn Glu Glu
850 855 860
Ile Trp Ala Leu His Gln Met Glu Cys Lys Leu Asp Arg Thr Lys Pro
865 870 875 880
Leu Ile Asp Arg Leu Ile Ala Ser Gly Trp Gly Leu Leu Lys Arg Gln
885 890 895
Met Ala Arg Leu Asp Ala Leu Lys Glu Leu Gly Trp Ile Pro Ala Pro
900 905 910
Asp Ser Ser Glu Asn Leu Ser Arg Glu Asp Gly Glu Ala Lys Asp Tyr
915 920 925
Arg Glu Ser Leu Ala Val Asp Asp Leu Met Phe Ser Ala Val Arg Thr
930 935 940
Leu Arg Leu Ala Leu Gln Arg His Gly Asn Arg Ala Arg Ile Ala Tyr
945 950 955 960
Tyr Leu Ile Ser Glu Val Lys Ile Arg Pro Gly Gly Ile Gln Glu Lys
965 970 975
Leu Asp Glu Asn Gly Arg Ile Asp Leu Leu Gln Asp Ala Leu Ala Leu
980 985 990
Trp His Glu Leu Phe Ser Ser Pro Gly Trp Arg Asp Glu Ala Ala Lys
995 1000 1005
Gln Leu Trp Asp Ser Arg Ile Ala Thr Leu Ala Gly Tyr Lys Ala
1010 1015 1020
Pro Glu Glu Asn Gly Asp Asn Val Ser Asp Val Ala Tyr Arg Lys
1025 1030 1035
Lys Gln Gln Val Tyr Arg Glu Gln Leu Arg Asn Val Ala Lys Thr
1040 1045 1050
Leu Ser Gly Asp Val Ile Thr Cys Lys Glu Leu Ser Asp Ala Trp
1055 1060 1065
Lys Glu Arg Trp Glu Asp Glu Asp Gln Arg Trp Lys Lys Leu Leu
1070 1075 1080
Arg Trp Phe Lys Asp Trp Val Leu Pro Ser Gly Thr Gln Ala Asn
1085 1090 1095
Asn Ala Thr Ile Arg Asn Val Gly Gly Leu Ser Leu Ser Arg Leu
1100 1105 1110
Ala Thr Ile Thr Glu Phe Arg Arg Lys Val Gln Val Gly Phe Phe
1115 1120 1125
Thr Arg Leu Arg Pro Asp Gly Thr Arg His Glu Ile Gly Glu Gln
1130 1135 1140
Phe Gly Gln Lys Thr Leu Asp Ala Leu Glu Leu Leu Arg Glu Gln
1145 1150 1155
Arg Val Lys Gln Leu Ala Ser Arg Ile Ala Glu Ala Ala Leu Gly
1160 1165 1170
Ile Gly Ser Glu Gly Gly Lys Gly Trp Asp Gly Gly Lys Arg Pro
1175 1180 1185
Arg Gln Arg Ile Asn Asp Ser Arg Phe Ala Pro Cys His Ala Val
1190 1195 1200
Val Ile Glu Asn Leu Ala Asn Tyr Arg Pro Asp Glu Thr Arg Thr
1205 1210 1215
Arg Leu Glu Asn Arg Arg Leu Met Thr Trp Ser Ala Ser Lys Val
1220 1225 1230
His Lys Tyr Leu Ser Glu Ala Cys Gln Leu Asn Gly Leu Tyr Leu
1235 1240 1245
Cys Thr Val Ser Ala Trp Tyr Thr Ser Arg Gln Asp Ser Arg Thr
1250 1255 1260
Gly Ala Pro Gly Ile Arg Cys Gln Asp Val Ser Val Arg Glu Phe
1265 1270 1275
Met Gln Ser Pro Phe Trp Arg Lys Gln Val Lys Gln Ala Glu Ala
1280 1285 1290
Lys His Asp Glu Asn Lys Gly Asp Ala Arg Glu Arg Phe Leu Cys
1295 1300 1305
Glu Leu Asn Lys Thr Trp Lys Ala Lys Thr Pro Ala Glu Trp Lys
1310 1315 1320
Lys Ala Gly Phe Val Arg Ile Pro Leu Arg Gly Gly Glu Ile Phe
1325 1330 1335
Val Ser Ala Asp Ser Lys Ser Pro Ser Ala Lys Gly Ile His Ala
1340 1345 1350
Asp Leu Asn Ala Ala Ala Asn Ile Gly Leu Arg Ala Leu Thr Asp
1355 1360 1365
Pro Asp Trp Pro Gly Lys Trp Trp Tyr Val Pro Cys Asp Pro Val
1370 1375 1380
Ser Phe Glu Ser Lys Met Asp Tyr Val Lys Gly Cys Ala Ala Val
1385 1390 1395
Lys Val Gly Gln Pro Leu Arg Gln Pro Ala Gln Thr Asn Ala Asp
1400 1405 1410
Gly Ala Ala Ser Lys Ile Arg Lys Gly Lys Lys Asn Arg Thr Ala
1415 1420 1425
Gly Thr Ser Lys Glu Lys Val Tyr Leu Trp Arg Asp Ile Ser Ala
1430 1435 1440
Phe Pro Leu Glu Ser Asn Glu Ile Gly Glu Trp Lys Glu Thr Ser
1445 1450 1455
Ala Tyr Gln Asn Asp Val Gln Tyr Arg Val Ile Arg Met Leu Lys
1460 1465 1470
Glu His Ile Lys Ser Leu Asp Asn Arg Thr Gly Asp Asn Val Glu
1475 1480 1485
Gly
<210> 558
<211> 277
<212> PRT
<213> Alicyclobacillus herbarius
<400> 558
Met Leu Lys Gln Ala Val Leu Gly Asn Gly Pro Leu Ile Asn Trp Glu
1 5 10 15
Lys Asn Val Lys Arg Gly Lys Gly Met Ala Thr Lys Ser Ile Lys Val
20 25 30
Lys Leu Arg Leu Gly Lys His Pro Asp Ile Arg Ala Gly Ile Trp Gln
35 40 45
Leu His Lys Ala Ala Asn Ala Gly Val Arg Tyr Tyr Thr Glu Trp Leu
50 55 60
Ser Leu Met Arg Gln Lys Asn Leu Tyr Thr Arg Gly Pro Lys Gly Glu
65 70 75 80
Gln Gln Leu Tyr Arg Ser Gly Glu Gln Cys Arg Arg Glu Leu Leu Gln
85 90 95
Arg Leu Arg Glu Arg Gln Arg Leu Asn Gly Arg Thr Asp Glu Pro Gly
100 105 110
Thr Asp Glu Glu Leu Leu Lys Val Ala Arg Gln Ile Tyr Glu Val Leu
115 120 125
Val Pro Gln Ser Ile Gly Lys Ser Gly Asp Ala Gln Gln Leu Ala Ser
130 135 140
Asn Phe Leu Ser Pro Leu Val Asp Pro Asn Ser Lys Gly Gly Gln Gly
145 150 155 160
Gln Ser Asn Ala Gly Arg Lys Pro Ala Trp Gln Lys Met Arg Asp Glu
165 170 175
Gly Asn Pro Gly Trp Val Ala Ala Lys Glu Arg Tyr Glu Gln Arg Lys
180 185 190
Ala Thr Asp Pro Thr Lys Lys Met Ile Glu Met Leu Asp Gly Leu Gly
195 200 205
Leu Lys Pro Leu Phe Ser Val Phe Thr Glu Thr Tyr Thr Thr Gly Val
210 215 220
Lys Trp Lys Asp Leu Ser Lys Arg Gln Gly Val Arg Thr Trp Asp Arg
225 230 235 240
Asp Met Phe Gln Ser Leu Ser Glu Arg Ser Gly Val Ile Asp Val Gly
245 250 255
Ser His Thr Val His His Ile Asp Leu Ala Thr Ala Ser Asp Ala Gln
260 265 270
Ile Gln Tyr Glu Leu
275
<210> 559
<211> 277
<212> PRT
<213> Alicyclobacillus contaminans
<400> 559
Met Ser Val Lys Ser Ile Lys Phe Lys Leu Met Ile Gly Gly Pro Gln
1 5 10 15
Tyr Thr Arg Ile Arg Arg Gly Ile Tyr Lys Thr His Glu Val Phe Asn
20 25 30
Glu Gly Val Arg Tyr Tyr Gln Glu Trp Leu Leu Leu Met Arg Gln Gly
35 40 45
Asp Val Tyr Arg Tyr Gln Asp Asp Lys Pro Glu Ile Val Leu Ser Ala
50 55 60
Glu His Cys Lys Arg Glu Leu Leu Arg Arg Leu Arg Gln Val Gln Lys
65 70 75 80
Glu Asn Val Gly Arg Thr Ser His Thr Asp Glu Glu Leu Leu Gln Val
85 90 95
Met Arg Ala Leu Tyr Glu Leu Ile Val Pro Ser Ala Val Gly Lys Lys
100 105 110
Gly Asp Ala Ala Ser Leu Ser Arg Lys Phe Leu Ser Pro Leu Ala Trp
115 120 125
Lys Asp Ser Lys Gly Leu Thr Gly Glu Ser Lys Ala Gly Asn Lys Pro
130 135 140
Arg Trp Lys Arg Leu Gln Glu Gln Gly Leu Pro Tyr Glu Glu Glu Tyr
145 150 155 160
Asn Arg Trp Leu Arg Glu Lys Glu Ser Asp Pro Ala Lys His Ile Pro
165 170 175
Ala Gln Leu Ala Ser Met Gly Leu Lys Pro Phe Leu Lys Val Phe Thr
180 185 190
Glu Ser Thr Glu Gly Ile Ala Trp Leu Pro Leu Ala Lys Asp Gln Gly
195 200 205
Val Arg Thr Trp Asp Arg Asp Met Phe Gln Gln Ala Ile Glu Gly Leu
210 215 220
Leu Ser Trp Glu Ser Trp Asn Arg Arg Val Arg Glu Glu Tyr Asp Ala
225 230 235 240
Leu Ser Ala Arg Val Tyr Ala Tyr His Ala Lys His Phe Ala Asp Gln
245 250 255
Pro Gly Trp Ala Val Tyr Trp Pro Gln Ser Gln Pro Arg Gln Lys Gly
260 265 270
Trp Val Lys Met Lys
275
<210> 560
<211> 218
<212> PRT
<213> Citrobacter freundii
<400> 560
Met Tyr Glu Leu Trp Ser Thr Phe Pro Ala Ser Ala Ser His Glu Gln
1 5 10 15
Tyr Lys Glu Ala Leu Lys Arg Val Gln Gln Arg Leu Arg Gly Arg Phe
20 25 30
Gly Asp Ala His Phe Phe Gln Tyr Leu Met Glu Glu Lys Asn Arg Leu
35 40 45
Ile Trp Lys Gly Asn Pro Gln Arg Ile His Tyr Phe Val Ala Arg Asn
50 55 60
Glu Leu Thr Lys Arg Leu Glu Glu Ala Lys Gln Ser Ala Thr Met Thr
65 70 75 80
Leu Pro Asn Ala Arg Lys His Pro Leu Trp Val Arg Phe Asp Ala Arg
85 90 95
Gly Gly Asn Leu Gln Asp Tyr Tyr Leu Thr Ala Glu Ala Asp Lys Pro
100 105 110
Arg Ser Arg Arg Phe Val Thr Phe Ser Gln Leu Ile Trp Pro Ser Glu
115 120 125
Ser Gly Trp Met Glu Lys Lys Asp Val Glu Val Glu Leu Ala Leu Ser
130 135 140
Arg Gln Phe Tyr Gln Gln Val Lys Leu Leu Lys Asn Asp Lys Gly Lys
145 150 155 160
Gln Lys Ile Glu Phe Lys Asp Lys Gly Ser Gly Ser Thr Phe Asn Gly
165 170 175
His Leu Gly Gly Ala Lys Leu Gln Leu Glu Arg Gly Asp Leu Glu Lys
180 185 190
Glu Glu Lys Thr Ser Arg Thr Gly Lys Ser Ala Ala Phe Thr Leu Thr
195 200 205
Leu Ser Leu Ile Ser Asn Leu Cys Lys Lys
210 215
<210> 561
<211> 218
<212> PRT
<213> Citrobacter freundii
<400> 561
Leu Tyr Glu Leu Trp Ser Thr Phe Pro Ala Ser Ala Ser His Glu Gln
1 5 10 15
Tyr Lys Glu Ala Leu Lys Arg Val Gln Gln Arg Leu Arg Gly Arg Phe
20 25 30
Gly Asp Ala His Phe Phe Gln Tyr Leu Met Glu Glu Lys Asn Arg Leu
35 40 45
Ile Trp Lys Gly Asn Pro Gln Arg Ile His Tyr Phe Val Ala Arg Asn
50 55 60
Glu Leu Thr Lys Arg Leu Glu Glu Ala Lys Gln Ser Ala Thr Met Thr
65 70 75 80
Leu Pro Asn Ala Arg Lys His Pro Leu Trp Val Arg Phe Asp Ala Arg
85 90 95
Gly Gly Asn Leu Gln Asp Tyr Tyr Leu Thr Ala Glu Ala Asp Lys Pro
100 105 110
Arg Ser Arg Arg Phe Val Thr Phe Ser Gln Leu Ile Trp Pro Ser Glu
115 120 125
Ser Gly Trp Met Glu Lys Lys Asp Val Glu Val Glu Leu Ala Leu Ser
130 135 140
Arg Gln Phe Tyr Gln Gln Val Lys Leu Leu Lys Asn Asp Lys Gly Lys
145 150 155 160
Gln Lys Ile Glu Phe Lys Asp Lys Gly Ser Gly Ser Thr Phe Asn Gly
165 170 175
His Leu Gly Gly Ala Lys Leu Gln Leu Glu Arg Gly Asp Leu Glu Lys
180 185 190
Glu Glu Lys Thr Ser Arg Thr Gly Lys Ser Ala Ala Phe Thr Leu Thr
195 200 205
Leu Ser Leu Ile Ser Asn Leu Cys Lys Lys
210 215
<210> 562
<211> 482
<212> PRT
<213> Brevibacillus agri
<400> 562
Met Glu Lys Arg Asp Glu Arg Phe Gln Leu His Gln Arg Val Lys Phe
1 5 10 15
Gln Ile Arg Val Leu Ala Gln Ile Met Arg Met Ala Asn Lys Gln Tyr
20 25 30
Gly Asp Arg Trp Asp Glu Leu Asp Ser Leu Lys Gln Ala Val Glu Gln
35 40 45
Lys Lys Ser Pro Leu Asp Gln Thr Asp Arg Thr Phe Trp Glu Gly Ile
50 55 60
Val Cys Asp Leu Thr Lys Val Leu Pro Arg Asn Glu Ala Asp Trp Glu
65 70 75 80
Gln Ala Val Val Gln Ile His Arg Lys Ala Glu Glu Tyr Val Gly Lys
85 90 95
Ala Val Gln Ala Trp Arg Lys Arg Phe Ala Ala Asp Glu Arg Lys Gly
100 105 110
Ile Ala Gly Leu Ser Met Trp Asn Ile Glu Glu Leu Glu Gly Leu Arg
115 120 125
Lys Leu Leu Ile Ser Trp Ser Arg Arg Thr Arg Asn Pro Gln Glu Val
130 135 140
Asn Arg Phe Glu Arg Gly His Thr Ser His Gln Arg Leu Leu Thr His
145 150 155 160
Ile Gln Asn Val Lys Glu Asp Arg Leu Lys Gln Leu Ser His Ala Ile
165 170 175
Val Met Thr Ala Leu Gly Tyr Val Tyr Asp Glu Arg Lys Gln Glu Trp
180 185 190
Cys Ala Glu Tyr Pro Ala Cys Gln Val Ile Leu Phe Glu Asn Leu Ser
195 200 205
Gln Tyr Arg Ser Asn Leu Asp Arg Ser Thr Lys Glu Asn Ser Thr Leu
210 215 220
Met Lys Trp Ala His Arg Ser Ile Pro Lys Tyr Val His Met Gln Ala
225 230 235 240
Glu Pro Tyr Gly Ile Gln Ile Gly Asp Val Arg Ala Glu Tyr Ser Ser
245 250 255
Arg Phe Tyr Ala Lys Thr Gly Thr Pro Gly Ile Arg Cys Lys Lys Val
260 265 270
Arg Gly Gln Asp Leu Gln Gly Arg Arg Phe Glu Asn Leu Gln Lys Arg
275 280 285
Leu Val Asn Glu Gln Phe Leu Thr Glu Glu Gln Val Lys Gln Leu Arg
290 295 300
Pro Gly Asp Ile Val Pro Asp Asp Ser Gly Glu Leu Phe Met Thr Leu
305 310 315 320
Thr Asp Gly Ser Gly Ser Lys Glu Val Val Phe Leu Gln Ala Asp Ile
325 330 335
Asn Ala Ala His Asn Leu Gln Lys Arg Phe Trp Gln Arg Tyr Asn Glu
340 345 350
Leu Phe Lys Val Ser Cys Arg Val Ile Val Arg Asp Glu Glu Glu Tyr
355 360 365
Leu Val Pro Lys Thr Lys Ser Val Gln Ala Lys Leu Gly Lys Gly Leu
370 375 380
Phe Val Lys Lys Ser Asp Thr Ala Trp Lys Asp Val Tyr Val Trp Asp
385 390 395 400
Ser Gln Ala Lys Leu Lys Gly Lys Thr Thr Phe Thr Glu Glu Ser Glu
405 410 415
Ser Pro Glu Gln Leu Glu Asp Phe Gln Glu Ile Ile Glu Glu Ala Glu
420 425 430
Glu Ala Lys Gly Thr Tyr Arg Thr Leu Phe Arg Asp Pro Ser Gly Val
435 440 445
Phe Phe Pro Glu Ser Val Trp Tyr Pro Gln Lys Asp Phe Trp Gly Glu
450 455 460
Val Lys Arg Lys Leu Tyr Gly Lys Leu Arg Glu Arg Phe Leu Thr Lys
465 470 475 480
Ala Arg
<210> 563
<211> 584
<212> PRT
<213> Brevibacillus agri
<400> 563
Met Ala Ile Arg Ser Ile Lys Leu Lys Leu Lys Thr His Thr Gly Pro
1 5 10 15
Glu Ala Gln Asn Leu Arg Lys Gly Ile Trp Arg Thr His Arg Leu Leu
20 25 30
Asn Glu Gly Val Ala Tyr Tyr Met Lys Met Leu Leu Leu Phe Arg Gln
35 40 45
Glu Ser Thr Gly Glu Arg Pro Lys Glu Glu Leu Gln Glu Glu Leu Ile
50 55 60
Cys His Ile Arg Glu Gln Gln Gln Arg Asn Gln Ala Asp Lys Asn Thr
65 70 75 80
Gln Ala Leu Pro Leu Asp Lys Ala Leu Glu Ala Leu Arg Gln Leu Tyr
85 90 95
Glu Leu Leu Val Pro Ser Ser Val Gly Gln Ser Gly Asp Ala Gln Ile
100 105 110
Ile Ser Arg Lys Phe Leu Ser Pro Leu Val Asp Pro Asn Ser Glu Gly
115 120 125
Gly Lys Gly Thr Ser Lys Ala Gly Ala Lys Pro Thr Trp Gln Lys Lys
130 135 140
Lys Glu Ala Asn Asp Pro Thr Trp Glu Gln Asp Tyr Glu Lys Trp Lys
145 150 155 160
Lys Arg Arg Glu Glu Asp Pro Thr Ala Ser Val Ile Thr Thr Leu Glu
165 170 175
Glu Tyr Gly Ile Arg Pro Ile Phe Pro Leu Tyr Thr Asn Thr Val Thr
180 185 190
Asp Ile Ala Trp Leu Pro Leu Gln Ser Asn Gln Phe Val Arg Thr Trp
195 200 205
Asp Arg Asp Met Leu Gln Gln Ala Ile Glu Arg Leu Leu Ser Trp Glu
210 215 220
Ser Trp Asn Lys Arg Val Gln Glu Glu Tyr Ala Lys Leu Lys Glu Lys
225 230 235 240
Met Ala Gln Leu Asn Glu Gln Leu Glu Gly Gly Gln Glu Trp Ile Ser
245 250 255
Leu Leu Glu Gln Tyr Glu Glu Asn Arg Glu Arg Glu Leu Arg Glu Asn
260 265 270
Met Thr Ala Ala Asn Asp Lys Tyr Arg Ile Thr Lys Arg Gln Met Lys
275 280 285
Gly Trp Asn Glu Leu Tyr Glu Leu Trp Ser Thr Phe Pro Ala Ser Ala
290 295 300
Ser His Glu Gln Tyr Lys Glu Ala Leu Lys Arg Val Gln Gln Arg Leu
305 310 315 320
Arg Gly Arg Phe Gly Asp Ala His Phe Phe Gln Tyr Leu Met Glu Glu
325 330 335
Lys Asn Arg Leu Ile Trp Lys Gly Asn Pro Gln Arg Ile His Tyr Phe
340 345 350
Val Ala Arg Asn Glu Leu Thr Lys Arg Leu Glu Glu Ala Lys Gln Ser
355 360 365
Ala Thr Met Thr Leu Pro Asn Ala Arg Lys His Pro Leu Trp Val Arg
370 375 380
Phe Asp Ala Arg Gly Gly Asn Leu Gln Asp Tyr Tyr Leu Thr Ala Glu
385 390 395 400
Ala Asp Lys Pro Arg Ser Arg Arg Phe Val Thr Phe Ser Gln Leu Ile
405 410 415
Trp Pro Ser Glu Ser Gly Trp Met Glu Lys Lys Asp Val Glu Val Glu
420 425 430
Leu Ala Leu Ser Arg Gln Phe Tyr Gln Gln Val Lys Leu Leu Lys Asn
435 440 445
Asp Lys Gly Lys Gln Lys Ile Glu Phe Lys Asp Lys Gly Ser Gly Ser
450 455 460
Thr Phe Asn Gly His Leu Gly Gly Ala Lys Leu Gln Leu Glu Arg Gly
465 470 475 480
Asp Leu Glu Lys Glu Glu Lys Asn Phe Glu Asp Gly Glu Ile Gly Ser
485 490 495
Val Tyr Leu Asn Val Val Ile Asp Phe Glu Pro Leu Gln Glu Val Lys
500 505 510
Asn Gly Arg Val Gln Ala Pro Tyr Gly Gln Val Leu Gln Leu Ile Arg
515 520 525
Arg Pro Asn Glu Phe Pro Lys Val Thr Thr Tyr Lys Ser Glu Gln Leu
530 535 540
Val Glu Trp Ile Lys Ala Ser Pro Gln His Ser Ala Gly Val Glu Ser
545 550 555 560
Leu Ala Ser Gly Phe Arg Val Met Ser Ile Asp Leu Gly Leu Arg Ala
565 570 575
Ala Ala Ala Thr Ser Ile Phe Leu
580
<210> 564
<211> 259
<212> PRT
<213> Brevibacillus sp.
<400> 564
Met Ala Ile Arg Ser Ile Lys Leu Lys Leu Lys Thr His Thr Gly Pro
1 5 10 15
Glu Ala Gln Asn Leu Arg Lys Gly Ile Trp Arg Thr His Arg Leu Leu
20 25 30
Asn Glu Gly Val Ala Tyr Tyr Met Lys Met Leu Leu Leu Phe Arg Gln
35 40 45
Glu Ser Thr Gly Glu Arg Pro Lys Glu Glu Leu Gln Glu Glu Leu Ile
50 55 60
Cys His Ile Arg Glu Gln Gln Gln Arg Asn Gln Ala Asp Lys Asn Thr
65 70 75 80
Gln Ala Leu Pro Leu Asp Lys Ala Leu Glu Ala Leu Arg Gln Leu Tyr
85 90 95
Glu Leu Leu Val Pro Ser Ser Val Gly Gln Ser Gly Asp Ala Gln Ile
100 105 110
Ile Ser Arg Lys Phe Leu Ser Pro Leu Val Asp Pro Asn Ser Glu Gly
115 120 125
Gly Lys Gly Thr Ser Lys Ala Gly Ala Lys Pro Thr Trp Gln Lys Lys
130 135 140
Lys Glu Ala Asn Asp Pro Thr Trp Glu Gln Asp Tyr Glu Lys Trp Lys
145 150 155 160
Lys Arg Arg Glu Glu Asp Pro Thr Ala Ser Val Ile Thr Thr Leu Glu
165 170 175
Glu Tyr Gly Ile Arg Pro Ile Phe Pro Leu Tyr Thr Asn Thr Val Thr
180 185 190
Asp Ile Ala Trp Leu Pro Leu Gln Ser Asn Gln Phe Val Arg Thr Trp
195 200 205
Asp Arg Asp Met Leu Gln Gln Ala Ile Glu Arg Leu Leu Ser Trp Glu
210 215 220
Ser Trp Asn Lys Arg Val Gln Glu Glu Tyr Ala Lys Leu Lys Glu Lys
225 230 235 240
Met Ala Gln Leu Asn Glu Gln Leu Glu Gly Gly Gln Glu Trp Cys Thr
245 250 255
Leu Ser Arg
<210> 565
<211> 658
<212> PRT
<213> Methylobacterium nodulans
<400> 565
Met Leu Thr Lys Gln Asp Lys Gln Gln Lys Ile Thr Tyr Cys Thr Asn
1 5 10 15
Met Asn Glu Val Phe Glu Ala Lys Leu Gly Ser Ala Asp Leu Leu Leu
20 25 30
Asn Trp Asp His Leu Arg Gly Arg Ile Arg Asp Arg Val Asp Ala Gly
35 40 45
Asp Ile Gly Ser Ala Phe Leu Lys Leu Ala Leu Asp Val Ala His Val
50 55 60
Leu Pro Asp Gly Val Asp Asp Gln Leu Ala Arg Ala Ala Phe His Phe
65 70 75 80
Gln Ser Ala Lys Gly Ala Lys Ser Lys His Ala Asp Ser Val Gln Ala
85 90 95
Gly Leu Arg Val Leu Ser Ile Asp Leu Gly Val Arg Ser Phe Ala Thr
100 105 110
Cys Ser Val Phe Glu Leu Lys Asp Thr Ala Pro Thr Thr Gly Val Ala
115 120 125
Phe Pro Leu Ala Glu Phe Arg Leu Trp Ala Val His Glu Arg Ser Phe
130 135 140
Thr Leu Glu Leu Pro Gly Glu Asn Val Gly Ala Ala Gly Gln Gln Trp
145 150 155 160
Arg Ala Gln Ala Asp Ala Glu Leu Arg Gln Leu Arg Gly Gly Leu Asn
165 170 175
Arg His Arg Gln Leu Leu Arg Ala Ala Thr Val Gln Lys Gly Glu Arg
180 185 190
Asp Ala Tyr Leu Thr Asp Leu Arg Glu Ala Trp Ser Ala Lys Glu Leu
195 200 205
Trp Pro Phe Glu Ala Ser Leu Leu Ser Glu Leu Glu Arg Cys Ser Thr
210 215 220
Val Ala Asp Pro Leu Trp Gln Asp Thr Cys Lys Arg Ala Ala Arg Leu
225 230 235 240
Tyr Arg Thr Glu Phe Gly Ala Val Val Ser Glu Trp Arg Ser Arg Thr
245 250 255
Arg Ser Arg Glu Asp Arg Lys Tyr Ala Gly Lys Ser Met Trp Ser Val
260 265 270
Gln His Leu Thr Asp Val Arg Arg Phe Leu Gln Ser Trp Ser Leu Ala
275 280 285
Gly Arg Ala Ser Gly Asp Ile Arg Arg Leu Asp Arg Glu Arg Gly Gly
290 295 300
Val Phe Ala Lys Asp Leu Leu Asp His Ile Asp Ala Leu Lys Asp Asp
305 310 315 320
Arg Leu Lys Thr Gly Ala Asp Leu Ile Val Gln Ala Ala Arg Gly Phe
325 330 335
Gln Arg Asn Glu Phe Gly Tyr Trp Val Gln Lys His Ala Pro Cys His
340 345 350
Val Ile Leu Phe Glu Asp Leu Ser Arg Tyr Arg Met Arg Thr Asp Arg
355 360 365
Pro Arg Arg Glu Asn Ser Gln Leu Met Gln Trp Ala His Arg Gly Val
370 375 380
Pro Asp Met Val Gly Met Gln Gly Glu Ile Tyr Gly Ile Gln Asp Arg
385 390 395 400
Arg Asp Pro Asp Ser Ala Arg Lys His Ala Arg Gln Pro Leu Ala Ala
405 410 415
Phe Cys Leu Asp Thr Pro Ala Ala Phe Ser Ser Arg Tyr His Ala Ser
420 425 430
Thr Met Thr Pro Gly Ile Arg Cys His Pro Leu Arg Lys Arg Glu Phe
435 440 445
Glu Asp Gln Gly Phe Leu Glu Leu Leu Lys Arg Glu Asn Glu Gly Leu
450 455 460
Asp Leu Asn Gly Tyr Lys Pro Gly Asp Leu Val Pro Leu Pro Gly Gly
465 470 475 480
Glu Val Phe Val Cys Leu Asn Ala Asn Gly Leu Ser Arg Ile His Ala
485 490 495
Asp Ile Asn Ala Ala Gln Asn Leu Gln Arg Arg Phe Trp Thr Gln His
500 505 510
Gly Asp Ala Phe Arg Leu Pro Cys Gly Lys Ser Ala Val Gln Gly Gln
515 520 525
Ile Arg Trp Ala Pro Leu Ser Met Gly Lys Arg Gln Ala Gly Ala Leu
530 535 540
Gly Gly Phe Gly Tyr Leu Glu Pro Thr Gly His Asp Ser Gly Ser Cys
545 550 555 560
Gln Trp Arg Lys Thr Thr Glu Ala Glu Trp Arg Arg Leu Ser Gly Ala
565 570 575
Gln Lys Asp Arg Asp Glu Ala Ala Ala Ala Glu Asp Glu Glu Leu Gln
580 585 590
Gly Leu Glu Glu Glu Leu Leu Glu Arg Ser Gly Glu Arg Val Val Phe
595 600 605
Phe Arg Asp Pro Ser Gly Val Val Leu Pro Thr Asp Leu Trp Phe Pro
610 615 620
Ser Ala Ala Phe Trp Ser Ile Val Arg Ala Lys Thr Val Gly Arg Leu
625 630 635 640
Arg Ser His Leu Asp Ala Gln Ala Glu Ala Ser Tyr Ala Val Ala Ala
645 650 655
Gly Leu
<210> 566
<211> 464
<212> PRT
<213> Methylobacterium nodulans
<400> 566
Met Pro Val Arg Ser Leu Lys Leu Lys Ile Val Val Pro Arg His Pro
1 5 10 15
Ser Glu Leu Glu Lys Ala Gln Ala Leu Trp Ser Thr His Arg Leu Val
20 25 30
Asn Glu Ala Val Ser Phe Tyr Glu Gln Lys Leu Leu Leu Leu Arg Gly
35 40 45
Glu Thr Tyr Ser Thr Ser Asp Gly Ser Val Pro Gln Asp Glu Val Arg
50 55 60
Arg Gln Leu Leu Glu Gln Ala Arg Glu Ala Gln Ala Arg Asn Gly Gly
65 70 75 80
Ser Gly Gly Ser Asp Asp Glu Ile Val Arg Leu Cys Arg Ser Leu Tyr
85 90 95
Glu Ala Ile Val Leu Ala Asp Asp Ala Asn Ala Gln Leu Ala Asn Ala
100 105 110
Phe Leu Gly Pro Leu Thr Asp Pro Asn Ser Ala Gly Phe Leu Glu Ala
115 120 125
Phe Asn Lys Val Asp Arg Pro Ala Pro Ser Trp Leu Asp Gln Val Pro
130 135 140
Ala Ser Asp Pro Ile Asp Pro Ala Val Leu Ala Glu Ala Asn Ala Trp
145 150 155 160
Leu Asp Thr Asp Ala Gly Arg Ala Trp Leu Val Asp Thr Gly Ala Pro
165 170 175
Pro Arg Trp Arg Ser Leu Ala Ala Lys Gln Asp Pro Ile Trp Pro Arg
180 185 190
Glu Phe Ala Arg Lys Leu Gly Glu Leu Arg Lys Glu Ala Ala Ser Gly
195 200 205
Thr Ser Ala Ile Ile Lys Ala Leu Lys Arg Asp Phe Gly Val Leu Pro
210 215 220
Leu Phe Gln Pro Ser Leu Ala Pro Arg Ile Leu Gly Ser Arg Ser Ser
225 230 235 240
Leu Thr Pro Trp Asp Arg Leu Ala Phe Arg Leu Ala Val Gly His Leu
245 250 255
Leu Ser Trp Glu Ser Trp Cys Thr Arg Ala Arg Asp Glu His Thr Ala
260 265 270
Arg Val Gln Arg Leu Glu Gln Phe Ser Ser Ala His Leu Lys Gly Asp
275 280 285
Leu Ala Thr Lys Val Ser Thr Leu Arg Glu Tyr Glu Arg Ala Arg Lys
290 295 300
Glu Gln Ile Ala Gln Leu Gly Leu Pro Met Gly Glu Arg Asp Phe Leu
305 310 315 320
Ile Thr Val Arg Met Thr Arg Gly Trp Asp Asp Leu Arg Glu Lys Trp
325 330 335
Arg Arg Ser Gly Asp Lys Gly Gln Glu Ala Leu His Ala Ile Ile Ala
340 345 350
Thr Glu Gln Thr Arg Lys Arg Gly Arg Phe Gly Asp Pro Asp Leu Phe
355 360 365
Arg Trp Leu Ala Arg Pro Glu Asn His His Val Trp Ala Asp Gly His
370 375 380
Ala Asp Ala Val Gly Val Leu Ala Arg Val Asn Ala Met Glu Arg Leu
385 390 395 400
Val Glu Arg Ser Arg Asp Thr Ala Leu Met Thr Leu Pro Asp Pro Val
405 410 415
Ala His Pro Arg Ser Ala Gln Trp Glu Ala Glu Gly Gly Ser Asn Leu
420 425 430
Arg Asn Tyr Gln Leu Glu Ala Val Gly Gly Glu Leu Gln Ile Thr Leu
435 440 445
Pro Leu Leu Lys Ala Ala Asp Asp Gly Arg Cys Ile Asp Thr Pro Leu
450 455 460
<210> 567
<211> 370
<212> PRT
<213> Methylobacterium nodulans
<400> 567
Met Tyr Glu Ala Ile Val Leu Ala Asp Asp Ala Asn Ala Gln Leu Ala
1 5 10 15
Asn Ala Phe Leu Gly Pro Leu Thr Asp Pro Asn Ser Ala Gly Phe Leu
20 25 30
Glu Ala Phe Asn Lys Val Asp Arg Pro Ala Pro Ser Trp Leu Asp Gln
35 40 45
Val Pro Ala Ser Asp Pro Ile Asp Pro Ala Val Leu Ala Glu Ala Asn
50 55 60
Ala Trp Leu Asp Thr Asp Ala Gly Arg Ala Trp Leu Val Asp Thr Gly
65 70 75 80
Ala Pro Pro Arg Trp Arg Ser Leu Ala Ala Lys Gln Asp Pro Ile Trp
85 90 95
Pro Arg Glu Phe Ala Arg Lys Leu Gly Glu Leu Arg Lys Glu Ala Ala
100 105 110
Ser Gly Thr Ser Ala Ile Ile Lys Ala Leu Lys Arg Asp Phe Gly Val
115 120 125
Leu Pro Leu Phe Gln Pro Ser Leu Ala Pro Arg Ile Leu Gly Ser Arg
130 135 140
Ser Ser Leu Thr Pro Trp Asp Arg Leu Ala Phe Arg Leu Ala Val Gly
145 150 155 160
His Leu Leu Ser Trp Glu Ser Trp Cys Thr Arg Ala Arg Asp Glu His
165 170 175
Thr Ala Arg Val Gln Arg Leu Glu Gln Phe Ser Ser Ala His Leu Lys
180 185 190
Gly Asp Leu Ala Thr Lys Val Ser Thr Leu Arg Glu Tyr Glu Arg Ala
195 200 205
Arg Lys Glu Gln Ile Ala Gln Leu Gly Leu Pro Met Gly Glu Arg Asp
210 215 220
Phe Leu Ile Thr Val Arg Met Thr Arg Gly Trp Asp Asp Leu Arg Glu
225 230 235 240
Lys Trp Arg Arg Ser Gly Asp Lys Gly Gln Glu Ala Leu His Ala Ile
245 250 255
Ile Ala Thr Glu Gln Thr Arg Lys Arg Gly Arg Phe Gly Asp Pro Asp
260 265 270
Leu Phe Arg Trp Leu Ala Arg Pro Glu Asn His His Val Trp Ala Asp
275 280 285
Gly His Ala Asp Ala Val Gly Val Leu Ala Arg Val Asn Ala Met Glu
290 295 300
Arg Leu Val Glu Arg Ser Arg Asp Thr Ala Leu Met Thr Leu Pro Asp
305 310 315 320
Pro Val Ala His Pro Arg Ser Ala Gln Trp Glu Ala Glu Gly Gly Ser
325 330 335
Asn Leu Arg Asn Tyr Gln Leu Glu Ala Val Gly Gly Glu Leu Gln Ile
340 345 350
Thr Leu Pro Leu Leu Lys Ala Ala Asp Asp Gly Arg Cys Ile Asp Thr
355 360 365
Pro Leu
370
<210> 568
<211> 1050
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(1050)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 568
Met Arg Ser Asn Tyr His Gly Gly Arg Asn Ala Arg Gln Trp Arg Lys
1 5 10 15
Gln Ile Ser Gly Leu Ala Arg Arg Thr Lys Glu Thr Val Phe Thr Tyr
20 25 30
Lys Phe Pro Leu Glu Thr Asp Ala Ala Glu Ile Asp Phe Asp Lys Ala
35 40 45
Val Gln Thr Tyr Gly Ile Ala Glu Gly Val Gly His Gly Ser Leu Ile
50 55 60
Gly Leu Val Cys Ala Phe His Leu Ser Gly Phe Arg Leu Phe Ser Lys
65 70 75 80
Ala Gly Glu Ala Met Ala Phe Arg Asn Arg Ser Arg Tyr Pro Thr Asp
85 90 95
Ala Phe Ala Glu Lys Leu Ser Ala Ile Met Gly Ile Gln Leu Pro Thr
100 105 110
Leu Ser Pro Glu Gly Leu Asp Leu Ile Phe Gln Ser Pro Pro Arg Ser
115 120 125
Arg Asp Gly Ile Ala Pro Val Trp Ser Glu Asn Glu Val Arg Asn Arg
130 135 140
Leu Tyr Thr Asn Trp Thr Gly Arg Gly Pro Ala Asn Lys Pro Asp Glu
145 150 155 160
His Leu Leu Glu Ile Ala Gly Glu Ile Ala Lys Gln Val Phe Pro Lys
165 170 175
Phe Gly Gly Trp Asp Asp Leu Ala Ser Asp Pro Asp Lys Ala Leu Ala
180 185 190
Ala Ala Asp Lys Tyr Phe Gln Ser Gln Gly Asp Phe Pro Ser Ile Ala
195 200 205
Ser Leu Pro Ala Ala Ile Met Leu Ser Pro Ala Asn Ser Thr Val Asp
210 215 220
Phe Glu Gly Asp Tyr Ile Ala Ile Asp Pro Ala Ala Glu Thr Leu Leu
225 230 235 240
His Gln Ala Val Ser Arg Cys Ala Ala Arg Leu Gly Arg Glu Arg Pro
245 250 255
Asp Leu Asp Gln Asn Lys Gly Pro Phe Val Ser Ser Leu Gln Asp Ala
260 265 270
Leu Val Ser Ser Gln Asn Asn Gly Leu Ser Trp Leu Phe Gly Val Gly
275 280 285
Phe Gln His Trp Lys Glu Lys Ser Pro Lys Glu Leu Ile Asp Glu Tyr
290 295 300
Lys Val Pro Ala Asp Gln His Gly Ala Val Thr Gln Val Lys Ser Phe
305 310 315 320
Val Asp Ala Ile Pro Leu Asn Pro Leu Phe Asp Thr Thr His Tyr Gly
325 330 335
Glu Phe Arg Ala Ser Val Ala Gly Lys Val Arg Ser Trp Val Ala Asn
340 345 350
Tyr Trp Lys Arg Leu Leu Asp Leu Lys Ser Leu Leu Ala Thr Thr Glu
355 360 365
Phe Thr Leu Pro Glu Ser Ile Ser Asp Pro Lys Ala Val Ser Leu Phe
370 375 380
Ser Gly Leu Leu Val Asp Pro Gln Gly Leu Lys Lys Val Ala Asp Ser
385 390 395 400
Leu Pro Ala Arg Leu Val Ser Ala Glu Glu Ala Ile Asp Arg Leu Met
405 410 415
Gly Val Gly Ile Pro Thr Ala Ala Asp Ile Ala Gln Val Glu Arg Val
420 425 430
Ala Asp Glu Ile Gly Ala Phe Ile Gly Gln Val Gln Gln Phe Asn Asn
435 440 445
Gln Val Lys Gln Lys Leu Glu Asn Leu Gln Asp Ala Asp Asp Glu Glu
450 455 460
Phe Leu Lys Gly Leu Lys Ile Glu Leu Pro Ser Gly Asp Lys Glu Pro
465 470 475 480
Pro Ala Ile Asn Arg Ile Ser Gly Gly Ala Pro Asp Ala Ala Ala Glu
485 490 495
Ile Ser Glu Leu Glu Glu Lys Leu Gln Arg Leu Leu Asp Ala Arg Ser
500 505 510
Glu His Phe Gln Thr Ile Ser Glu Trp Ala Glu Glu Asn Ala Val Thr
515 520 525
Leu Asp Pro Ile Ala Ala Met Val Glu Leu Glu Arg Leu Arg Leu Ala
530 535 540
Glu Arg Gly Ala Thr Gly Asp Pro Glu Glu Tyr Ala Leu Arg Leu Leu
545 550 555 560
Leu Gln Arg Ile Gly Arg Leu Ala Asn Arg Val Ser Pro Val Ser Ala
565 570 575
Gly Ser Ile Arg Glu Leu Leu Lys Pro Val Phe Met Glu Glu Arg Glu
580 585 590
Phe Asn Leu Phe Phe His Asn Arg Leu Gly Ser Leu Tyr Arg Ser Pro
595 600 605
Tyr Ser Thr Ser Arg His Gln Pro Phe Ser Ile Asp Val Gly Lys Ala
610 615 620
Lys Ala Ile Asp Trp Ile Ala Gly Leu Asp Gln Ile Ser Ser Asp Ile
625 630 635 640
Glu Lys Ala Leu Ser Gly Ala Gly Glu Ala Leu Gly Asp Gln Leu Arg
645 650 655
Asp Trp Ile Asn Leu Ala Gly Phe Ala Ile Ser Gln Arg Leu Arg Gly
660 665 670
Leu Pro Asp Thr Val Pro Asn Ala Leu Ala Gln Val Arg Cys Pro Asp
675 680 685
Asp Val Arg Ile Pro Pro Leu Leu Ala Met Leu Leu Glu Glu Asp Asp
690 695 700
Ile Ala Arg Asp Val Cys Leu Lys Ala Phe Asn Leu Tyr Val Ser Ala
705 710 715 720
Ile Asn Gly Cys Leu Phe Gly Ala Leu Arg Glu Gly Phe Ile Val Arg
725 730 735
Thr Arg Phe Gln Arg Ile Gly Thr Asp Gln Ile His Tyr Val Pro Lys
740 745 750
Asp Lys Ala Trp Glu Tyr Pro Asp Arg Leu Asn Thr Ala Lys Gly Pro
755 760 765
Ile Asn Ala Ala Val Ser Ser Asp Trp Ile Glu Lys Asp Gly Ala Val
770 775 780
Ile Lys Pro Val Glu Thr Val Arg Asn Leu Ser Ser Thr Gly Phe Ala
785 790 795 800
Gly Ala Gly Val Ser Glu Tyr Leu Val Gln Ala Pro His Asp Trp Tyr
805 810 815
Thr Pro Leu Asp Leu Arg Asp Val Ala His Leu Val Thr Gly Leu Pro
820 825 830
Val Glu Lys Asn Ile Thr Lys Leu Lys Arg Leu Thr Asn Arg Thr Ala
835 840 845
Phe Arg Met Val Gly Ala Ser Ser Phe Lys Thr His Leu Asp Ser Val
850 855 860
Leu Leu Ser Asp Lys Ile Lys Leu Gly Asp Phe Thr Ile Ile Ile Asp
865 870 875 880
Gln His Tyr Arg Gln Ser Val Thr Tyr Gly Gly Lys Val Lys Ile Ser
885 890 895
Tyr Glu Pro Glu Arg Leu Gln Val Glu Ala Ala Val Pro Val Val Asp
900 905 910
Thr Arg Asp Arg Thr Val Pro Glu Pro Asp Thr Leu Phe Asp His Ile
915 920 925
Val Ala Ile Asp Leu Gly Glu Arg Ser Val Gly Phe Ala Val Phe Asp
930 935 940
Ile Lys Ser Cys Leu Arg Thr Gly Glu Val Lys Pro Ile His Asp Asn
945 950 955 960
Asn Gly Asn Pro Val Val Gly Thr Val Ala Val Pro Ser Ile Arg Arg
965 970 975
Leu Met Lys Ala Val Arg Ser His Arg Arg Arg Arg Gln Pro Asn Gln
980 985 990
Lys Val Asn Gln Thr Tyr Ser Thr Ala Leu Gln Asn Tyr Arg Glu Asn
995 1000 1005
Val Ile Gly Asp Val Cys Asn Arg Ile Asp Thr Leu Met Glu Arg
1010 1015 1020
Tyr Asn Ala Phe Pro Val Leu Glu Phe Gln Ile Lys Asn Phe Gln
1025 1030 1035
Ala Gly Ala Lys Gln Leu Glu Ile Val Tyr Gly Ser
1040 1045 1050
<210> 569
<211> 1222
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(1222)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 569
Met Lys Lys Phe Glu Leu Lys Gln Asn Phe Arg Asn Asn Tyr Ser Gly
1 5 10 15
Lys Thr Leu Arg Asn Phe Arg Gln Thr Leu Ala Gln Ile Ala Asn Lys
20 25 30
Lys Ser Ser Asp Ser Ile Leu Thr Ile Lys Phe Lys Leu Asp Cys Ser
35 40 45
Lys Thr Gly Lys Leu Pro Lys Tyr Glu Asn Leu Ile Ser Leu Tyr Asp
50 55 60
Thr Ile Glu Asp Ile Lys Lys Gly Thr Leu Ser Tyr Tyr Leu Phe Thr
65 70 75 80
Leu Ile Val Ser Gly Phe Lys Phe Phe Gly Ser Ala Ser Gln Ala Lys
85 90 95
Ala Phe Ser Thr Lys Asp Ile Phe Lys Asp Asn Asp Phe Tyr Asn Gln
100 105 110
Phe Lys Ile Gln Ser His Leu Asp Leu Pro Asp Phe Val Pro Ser Lys
115 120 125
Ile Tyr Gln Arg Leu Lys Lys Asn Val Arg Ser Thr Asn Gly Lys Asp
130 135 140
Asn Ala Phe Lys Ala Ser Val Ile Val Ala Glu Tyr Arg Lys Glu Ile
145 150 155 160
Gly Lys Leu Lys Asn Lys Asp Glu Ser Ser Glu His Gln Cys Glu Glu
165 170 175
Leu Phe Lys Lys Ile Gly Thr Ala Leu Glu Thr Arg Phe Ser Ser Trp
180 185 190
Gln Asp Leu Ile Asn Asn Cys Ser Thr Gly Cys Glu Ile Ile Asp Glu
195 200 205
Ile Leu Asn Asp Ser Phe Gly Thr Leu Pro Ser Ile Lys Lys Met Val
210 215 220
Leu Ala Ser Thr Thr Gln Ser Ser Asp Gly Glu Gln Asp Gly Ile Ala
225 230 235 240
Ile Ala Tyr Asp Pro Asp Ser Thr Phe Ile Lys Ser Asp Glu Leu Leu
245 250 255
Asn Pro Tyr Phe Ala Val Ala Thr Ile Leu Lys Ser Met Pro Pro Glu
260 265 270
Ile Gln Gln Asp Lys Lys Ser Ala Tyr Val Lys Ala Asn Leu Thr Thr
275 280 285
Pro Thr His Asn Ala Leu Ser Trp Ile Phe Gly Lys Gly Leu Thr Leu
290 295 300
Phe Gln Thr Glu Ser Thr Glu Lys Leu Cys Ala Met Phe Asn Val Ser
305 310 315 320
Asp Lys Arg Val Ile Glu Gln Val Gln Asp Ala Ala Lys Ala Val Lys
325 330 335
Leu Pro Ala Glu Leu Asp Leu Asn His Cys Thr Leu Lys Phe Gln Asp
340 345 350
Phe Arg Ser Ser Leu Gly Gly His Leu Asp Ser Trp Thr Thr Asn Tyr
355 360 365
Leu Lys Arg Leu Asp Glu Leu Asn Asp Leu Leu Leu Asn Leu Pro Lys
370 375 380
Asn Leu Ser Leu Pro Asp Ile Phe Met Ile Asp Gly Lys Asp Phe Ile
385 390 395 400
Glu Tyr Ser Gly Cys Asn Arg Asp Glu Ile Gln Gln Met Ile Asp Phe
405 410 415
Val Val Asn Glu Gln Asn Arg Ile Lys Leu Gln Glu Ser Leu Asn Ala
420 425 430
Leu Leu Gly Lys Gly Asn Asn Gln Ile Cys Ser Asp Asp Ile Ser Thr
435 440 445
Val Lys Asp Phe Ser Glu Ile Val Asn Ser Leu His Ser Phe Val Gln
450 455 460
Gln Ile Asp Asn Ser Leu Glu Gln Ser Ser Asn Glu Ala Asn Ser Ile
465 470 475 480
Phe Ser Glu Leu Lys Lys Lys Ile Glu Lys Asn Glu Lys Trp Asp Ile
485 490 495
Trp Lys Asn Asn Leu Lys Lys Ile Pro Lys Leu Asn Lys Leu Ser Gly
500 505 510
Gly Val Pro Asp Ala Trp Lys Glu Ile Arg Glu Ile Glu Gln Lys Phe
515 520 525
His Glu Ile Ser Glu Asn Gln Lys Lys His Phe Thr Glu Val Met Glu
530 535 540
Trp Ile Asp Ala Gly Asn Gly Thr Ile Asp Ile Phe Glu Ser Arg Phe
545 550 555 560
Lys Tyr Asp Glu Leu Leu Lys Lys Ser Lys Lys Asn Asn Leu Gln Ser
565 570 575
Ala Asp Glu Leu Ala Phe Arg Ser Val Leu Asn Lys Leu Gly Arg Phe
580 585 590
Ala Arg Gln Gly Asn Asp Leu Val Cys Glu Lys Ile Lys Asn Trp Phe
595 600 605
Lys Glu Gln Asn Ile Phe Asp Ser Ser Lys Asp Phe Asn Arg Tyr Phe
610 615 620
Ile Asn Gln Lys Gly Phe Ile Phe Lys His Pro Ser Ser Lys Lys Asp
625 630 635 640
Asn Ser Pro Tyr Asn Leu Ser Ala Asn Leu Leu Glu Lys Arg Tyr Glu
645 650 655
Val Thr Asn Thr Val Gly Ala Leu Leu Glu Gln Cys Glu Ser Asp Pro
660 665 670
Ala Ile Val Asn Asp Pro Phe Ser Met Arg Ser Leu Val Glu Phe Arg
675 680 685
Ala Leu Trp Phe Ser Ile Asn Ile Ser Gly Ile Ser Lys Glu Gln His
690 695 700
Ile Pro Thr Lys Ile Ala Gln Pro Lys Leu Asp Asp Ser Thr Tyr Gln
705 710 715 720
Glu Ser Val Ser Pro Thr Leu Lys Tyr Arg Leu Glu Lys Glu Gln Ile
725 730 735
Thr Ser Ser Glu Leu Asn Ser Ile Phe Thr Val Tyr Lys Ser Leu Leu
740 745 750
Ser Gly Leu Ser Ile Arg Leu Ser Arg Asn Ser Phe Tyr Leu Arg Thr
755 760 765
Lys Phe Ser Trp Ile Gly Asn Asn Ser Leu Ile Tyr Cys Pro Lys Glu
770 775 780
Thr Thr Trp Lys Ile Pro Ala Ala Tyr Phe Lys Ser Asp Leu Trp Asn
785 790 795 800
Glu Tyr Lys Asp Lys Gln Ile Leu Ile Val Asn Glu Glu Tyr Asp Val
805 810 815
Asp Val Val Lys Thr Phe Glu Ser Val Tyr Lys Ile Val Lys Ser Lys
820 825 830
Asp Asn Asn Glu Lys Asn Arg Ile Leu Pro Leu Leu Lys Gln Leu Pro
835 840 845
His Asp Trp Met Phe Lys Leu Pro Phe Gly Ala Ser Asn Ala Glu Lys
850 855 860
Cys Lys Val Leu Lys Leu Glu Lys Asn Asn Lys Lys Phe Lys Pro Leu
865 870 875 880
Ser Val Ser Lys Asp Ser Leu Ala Arg Leu Ser Gly Pro Ser Thr Tyr
885 890 895
Phe Asn Gln Ile Asp Glu Ile Met Met Asn Asp Glu Ser Glu Leu Ser
900 905 910
Glu Met Thr Leu Leu Ala Asp Glu Pro Val Arg Gln Gln Met Ser Asn
915 920 925
Gly Lys Ile Glu Ile Ile Pro Asp Asp Tyr Val Met Ser Leu Ala Ile
930 935 940
Pro Ile Thr Arg Ser Leu Lys Lys Gly Asn Thr Glu Ser Phe Pro Phe
945 950 955 960
Lys Asn Ile Val Ser Ile Asp Gln Gly Glu Ala Gly Phe Ala Tyr Ala
965 970 975
Val Phe Lys Leu Ser Asp Cys Gly Asn Glu Arg Ala Glu Pro Ile Ala
980 985 990
Thr Gly Leu Ile Pro Ile Pro Ser Ile Arg Arg Leu Ile His Ser Val
995 1000 1005
Lys Lys Tyr Arg Gly Lys Lys Gln Arg Ile Gln Asn Phe Asn Gln
1010 1015 1020
Lys Phe Asp Ser Thr Met Phe Thr Leu Arg Glu Asn Val Thr Gly
1025 1030 1035
Asp Ile Cys Gly Leu Ile Val Ala Leu Met Lys Lys Tyr Asn Ala
1040 1045 1050
Phe Pro Ile Leu Glu Lys Gln Val Gly Asn Leu Glu Ser Gly Ser
1055 1060 1065
Lys Gln Leu Met Leu Val Tyr Lys Ala Val Asn Ser Lys Phe Leu
1070 1075 1080
Ala Ala Lys Val Asp Met Gln Asn Asp Gln Arg Arg Ser Trp Trp
1085 1090 1095
Tyr Gln Gly Asn Ser Trp Asn Thr Pro Ile Leu Arg Ile Ser Asn
1100 1105 1110
Pro Asn Gln Ser Asn Asn Lys Asn Ile Val Lys Asn Ile Asn Gly
1115 1120 1125
Lys Lys Tyr Glu Glu Leu Lys Ile Tyr Pro Gly Tyr Ser Val Ser
1130 1135 1140
Ala Tyr Met Thr Ser Cys Ile Cys His Val Cys Gly Arg Asn Ala
1145 1150 1155
Leu Glu Leu Leu Lys Asn Asp Asp Ser Thr Gly Lys Val Lys Lys
1160 1165 1170
Tyr Gln Ile Asn Gln Asp Gly Glu Val Thr Ile Gly Gly Glu Val
1175 1180 1185
Ile Lys Leu Tyr Arg Lys Pro Asp Arg Leu Thr Pro Val Lys Asn
1190 1195 1200
Leu Ala Lys Lys Gly Asn Arg Glu Arg Thr Tyr Ala Ser Ile Asn
1205 1210 1215
Glu Arg Ala Pro
1220
<210> 570
<211> 1255
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(1255)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 570
Met Arg Ser Asn Tyr His Gly Gly Arg Asn Ala Arg Gln Trp Arg Lys
1 5 10 15
Gln Ile Ser Gly Leu Ala Arg Arg Thr Lys Glu Thr Val Phe Thr Tyr
20 25 30
Lys Phe Pro Leu Glu Thr Asp Ala Ala Glu Ile Asp Phe Asp Lys Ala
35 40 45
Val Gln Thr Tyr Gly Ile Ala Glu Gly Val Gly His Gly Ser Leu Ile
50 55 60
Gly Leu Val Cys Ala Phe His Leu Ser Gly Phe Arg Leu Phe Ser Lys
65 70 75 80
Ala Gly Glu Ala Met Ala Phe Arg Asn Arg Ser Arg Tyr Pro Thr Asp
85 90 95
Ala Phe Ala Glu Lys Leu Ser Ala Ile Met Gly Ile Gln Leu Pro Thr
100 105 110
Leu Ser Pro Glu Gly Leu Asp Leu Ile Phe Gln Ser Pro Pro Arg Ser
115 120 125
Arg Asp Gly Ile Ala Pro Val Trp Ser Glu Asn Glu Val Arg Asn Arg
130 135 140
Leu Tyr Thr Asn Trp Thr Gly Arg Gly Pro Ala Asn Lys Pro Asp Glu
145 150 155 160
His Leu Leu Glu Ile Ala Gly Glu Ile Ala Lys Gln Val Phe Pro Lys
165 170 175
Phe Gly Gly Trp Asp Asp Leu Ala Ser Asp Pro Asp Lys Ala Leu Ala
180 185 190
Ala Ala Asp Lys Tyr Phe Gln Ser Gln Gly Asp Phe Pro Ser Ile Ala
195 200 205
Ser Leu Pro Ala Ala Ile Met Leu Ser Pro Ala Asn Ser Thr Val Asp
210 215 220
Phe Glu Gly Asp Tyr Ile Ala Ile Asp Pro Ala Ala Glu Thr Leu Leu
225 230 235 240
His Gln Ala Val Ser Arg Cys Ala Ala Arg Leu Gly Arg Glu Arg Pro
245 250 255
Asp Leu Asp Gln Asn Lys Gly Pro Phe Val Ser Ser Leu Gln Asp Ala
260 265 270
Leu Val Ser Ser Gln Asn Asn Gly Leu Ser Trp Leu Phe Gly Val Gly
275 280 285
Phe Gln His Trp Lys Glu Lys Ser Pro Lys Glu Leu Ile Asp Glu Tyr
290 295 300
Lys Val Pro Ala Asp Gln His Gly Ala Val Thr Gln Val Lys Ser Phe
305 310 315 320
Val Asp Ala Ile Pro Leu Asn Pro Leu Phe Asp Thr Thr His Tyr Gly
325 330 335
Glu Phe Arg Ala Ser Val Ala Gly Lys Val Arg Ser Trp Val Ala Asn
340 345 350
Tyr Trp Lys Arg Leu Leu Asp Leu Lys Ser Leu Leu Ala Thr Thr Glu
355 360 365
Phe Thr Leu Pro Glu Ser Ile Ser Asp Pro Lys Ala Val Ser Leu Phe
370 375 380
Ser Gly Leu Leu Val Asp Pro Gln Gly Leu Lys Lys Val Ala Asp Ser
385 390 395 400
Leu Pro Ala Arg Leu Val Ser Ala Glu Glu Ala Ile Asp Arg Leu Met
405 410 415
Gly Val Gly Ile Pro Thr Ala Ala Asp Ile Ala Gln Val Glu Arg Val
420 425 430
Ala Asp Glu Ile Gly Ala Phe Ile Gly Gln Val Gln Gln Phe Asn Asn
435 440 445
Gln Val Lys Gln Lys Leu Glu Asn Leu Gln Asp Ala Asp Asp Glu Glu
450 455 460
Phe Leu Lys Gly Leu Lys Ile Glu Leu Pro Ser Gly Asp Lys Glu Pro
465 470 475 480
Pro Ala Ile Asn Arg Ile Ser Gly Gly Ala Pro Asp Ala Ala Ala Glu
485 490 495
Ile Ser Glu Leu Glu Glu Lys Leu Gln Arg Leu Leu Asp Ala Arg Ser
500 505 510
Glu His Phe Gln Thr Ile Ser Glu Trp Ala Glu Glu Asn Ala Val Thr
515 520 525
Leu Asp Pro Ile Ala Ala Met Val Glu Leu Glu Arg Leu Arg Leu Ala
530 535 540
Glu Arg Gly Ala Thr Gly Asp Pro Glu Glu Tyr Ala Leu Arg Leu Leu
545 550 555 560
Leu Gln Arg Ile Gly Arg Leu Ala Asn Arg Val Ser Pro Val Ser Ala
565 570 575
Gly Ser Ile Arg Glu Leu Leu Lys Pro Val Phe Met Glu Glu Arg Glu
580 585 590
Phe Asn Leu Phe Phe His Asn Arg Leu Gly Ser Leu Tyr Arg Ser Pro
595 600 605
Tyr Ser Thr Ser Arg His Gln Pro Phe Ser Ile Asp Val Gly Lys Ala
610 615 620
Lys Ala Ile Asp Trp Ile Ala Gly Leu Asp Gln Ile Ser Ser Asp Ile
625 630 635 640
Glu Lys Ala Leu Ser Gly Ala Gly Glu Ala Leu Gly Asp Gln Leu Arg
645 650 655
Asp Trp Ile Asn Leu Ala Gly Phe Ala Ile Ser Gln Arg Leu Arg Gly
660 665 670
Leu Pro Asp Thr Val Pro Asn Ala Leu Ala Gln Val Arg Cys Pro Asp
675 680 685
Asp Val Arg Ile Pro Pro Leu Leu Ala Met Leu Leu Glu Glu Asp Asp
690 695 700
Ile Ala Arg Asp Val Cys Leu Lys Ala Phe Asn Leu Tyr Val Ser Ala
705 710 715 720
Ile Asn Gly Cys Leu Phe Gly Ala Leu Arg Glu Gly Phe Ile Val Arg
725 730 735
Thr Arg Phe Gln Arg Ile Gly Thr Asp Gln Ile His Tyr Val Pro Lys
740 745 750
Asp Lys Ala Trp Glu Tyr Pro Asp Arg Leu Asn Thr Ala Lys Gly Pro
755 760 765
Ile Asn Ala Ala Val Ser Ser Asp Trp Ile Glu Lys Asp Gly Ala Val
770 775 780
Ile Lys Pro Val Glu Thr Val Arg Asn Leu Ser Ser Thr Gly Phe Ala
785 790 795 800
Gly Ala Gly Val Ser Glu Tyr Leu Val Gln Ala Pro His Asp Trp Tyr
805 810 815
Thr Pro Leu Asp Leu Arg Asp Val Ala His Leu Val Thr Gly Leu Pro
820 825 830
Val Glu Lys Asn Ile Thr Lys Leu Lys Arg Leu Thr Asn Arg Thr Ala
835 840 845
Phe Arg Met Val Gly Ala Ser Ser Phe Lys Thr His Leu Asp Ser Val
850 855 860
Leu Leu Ser Asp Lys Ile Lys Leu Gly Asp Phe Thr Ile Ile Ile Asp
865 870 875 880
Gln His Tyr Arg Gln Ser Val Thr Tyr Gly Gly Lys Val Lys Ile Ser
885 890 895
Tyr Glu Pro Glu Arg Leu Gln Val Glu Ala Ala Val Pro Val Val Asp
900 905 910
Thr Arg Asp Arg Thr Val Pro Glu Pro Asp Thr Leu Phe Asp His Ile
915 920 925
Val Ala Ile Asp Leu Gly Glu Arg Ser Val Gly Phe Ala Val Phe Asp
930 935 940
Ile Lys Ser Cys Leu Arg Thr Gly Glu Val Lys Pro Ile His Asp Asn
945 950 955 960
Asn Gly Asn Pro Val Val Gly Thr Val Ala Val Pro Ser Ile Arg Arg
965 970 975
Leu Met Lys Ala Val Arg Ser His Arg Arg Arg Arg Gln Pro Asn Gln
980 985 990
Lys Val Asn Gln Thr Tyr Ser Thr Ala Leu Gln Asn Tyr Arg Glu Asn
995 1000 1005
Val Ile Gly Asp Val Cys Asn Arg Ile Asp Thr Leu Met Glu Arg
1010 1015 1020
Tyr Asn Ala Phe Pro Val Leu Glu Phe Gln Ile Lys Asn Phe Gln
1025 1030 1035
Ala Gly Ala Lys Gln Leu Glu Ile Val Tyr Gly Ser Val Leu His
1040 1045 1050
Arg Tyr Thr Phe Ser Gly Val Asp Ala His Lys Ala Lys Arg Arg
1055 1060 1065
Glu Tyr Trp Tyr Asn Gly Glu Leu Trp Glu His Pro Tyr Leu Met
1070 1075 1080
Ala Lys Lys Trp Asn Glu Glu Thr Asn Ser Met Ser Gly Ala Pro
1085 1090 1095
Lys Pro Val Ser Leu Phe Pro Gly Val Thr Val Asn Ala Ala Arg
1100 1105 1110
Thr Ser Gln Ile Cys His Gln Cys Gln Arg Asn Pro Met Ser His
1115 1120 1125
Leu Arg Gly Leu Thr Gly Thr Ile Glu Ile Ser Ser Asp Gly Leu
1130 1135 1140
Leu Glu Leu Asp Asp Gly Thr Ile Arg Leu Phe Glu Thr Ser Asp
1145 1150 1155
Tyr Asp Glu Asp Lys Phe Lys Gln Ser Arg Arg Glu Lys Arg Arg
1160 1165 1170
Leu Asp Ala Asn Val Leu Leu Ser Gly Arg His Arg Ala Glu Tyr
1175 1180 1185
Ile Tyr Thr Val Ala Lys Arg Asn Leu Arg Arg Pro Pro Lys Asn
1190 1195 1200
Val Met Thr Lys Asp Thr Thr Gln Ser Arg Tyr Thr Cys Leu Tyr
1205 1210 1215
Lys Asn Cys Ser Trp Thr Gly His Ala Asp Glu Asn Ala Ala Ile
1220 1225 1230
Asn Ile Gly Arg Arg Tyr Leu Ala Glu Arg Ile Asp Met Pro Ala
1235 1240 1245
Ser Lys Thr Lys Ala Ala Val
1250 1255
<210> 571
<211> 1258
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(1258)
<223> /note="Description of Unknown: Metagenome sequence"
<400> 571
Met Asn Ala Arg Asp Trp Arg Lys His Val Gly Val Leu Ala Gln Gln
1 5 10 15
His Lys Glu Thr Thr Arg Thr Tyr Thr Phe Pro Leu Asp Thr Thr Gly
20 25 30
Ser Ala Ile Asp Phe Asp Ala Ala Leu Gln Ala Tyr Asn Ala Val Glu
35 40 45
Gly Val Gly Tyr Gly Ser Leu Leu Gly Leu Ala Cys Ala Val His Leu
50 55 60
Ser Gly Phe Arg Leu Phe Ser Thr Gly Lys Glu Ala Ala Thr Phe Arg
65 70 75 80
Asn Arg Ala Arg Tyr Pro Asn Ala Ala Phe Gln Ala Ala Leu Arg Lys
85 90 95
Glu Leu Gly Thr Thr Ile Thr Thr Leu Thr Pro Glu Thr Leu Asp Arg
100 105 110
Leu Phe Ser Ser Arg Pro Lys Arg Arg Asn Gly Val Pro Leu Pro Trp
115 120 125
Asn Gln Asp Ser Ile Arg Asp Arg Leu Tyr Thr Asn Trp Val Lys Pro
130 135 140
Arg Pro Gly Asp Thr Pro Asp Ala Val Leu Phe Gln Ile Ala Thr Gly
145 150 155 160
Ile Ala Gln Glu Ile Thr Glu Asp Val Ser Ser Trp Thr Asp Leu Ala
165 170 175
Lys Asn Ser Asp Arg Gly Leu Lys Ala Ala His Arg Tyr Phe Ala Arg
180 185 190
Val Gly Gly Phe Pro Ala Phe Asp Asn Leu Thr Pro Pro Ala Thr Val
195 200 205
Gln Pro Thr Asp Thr Thr Ile Asp Tyr Asp Pro Asn Ala Pro Phe His
210 215 220
Leu Val Ser His Ala Asp Gln Thr Leu Ile His Gln Ser Ile Ser Leu
225 230 235 240
Cys Ala His Arg Ile Arg Gln Glu Asp Pro Ala Leu Asp Pro Asn Lys
245 250 255
Ser Gly Phe Ile Lys Gln Leu Gln Asn Asn Phe Leu Ser Gln Thr Phe
260 265 270
Tyr Gly Leu Ser Trp Leu Phe Gly Ala Gly Tyr Val His Phe Arg Glu
275 280 285
Cys Thr Ala Asn Asp Leu Ala Ile Gln Tyr Gly Ile Pro Asn Asn Cys
290 295 300
Arg Asp Gly Ile His Gln Ile Lys Ser Phe Ala Asp Ala Ile Leu Pro
305 310 315 320
Asn Thr Phe Phe Glu Lys Lys His Tyr Arg Lys Asp Ser Arg Ser Val
325 330 335
Gly Lys Lys Ala Lys Ser Trp Ile Ser Asn Tyr Trp Gln Arg Leu Leu
340 345 350
Gln Leu Gln Thr Trp Val Asp Asp His Thr Trp Val Thr Leu Pro Gln
355 360 365
Glu Leu Thr Glu Ala Gln Phe Lys Pro Leu Phe Arg Gly Leu Leu Val
370 375 380
Asp Ala Val Glu Leu Met Ala Ile Ala Glu Arg Leu Pro Gln Arg Leu
385 390 395 400
Ala Asp Cys Arg Asp Ser Leu Asp Cys Leu Met Gly Lys Gly Pro Gln
405 410 415
Ala Ala Thr Lys Asn Asp Val Glu Ile Val Glu Lys Val Arg Glu Glu
420 425 430
Ile Glu Ser Phe Val Gly Gln Ile Glu Gln Leu Gly Asn Gln Leu Arg
435 440 445
His Gln Leu Glu Asn Glu Asn Asn Asp Gln Val His Arg Asp Asn Leu
450 455 460
His Gln Leu Lys Asn Arg Leu Pro Leu Asp Leu Arg Arg Pro Gln Ala
465 470 475 480
Leu Asn Lys Ile Ser Gly Gly Val Pro Asp Val Ala Lys Ser Ile Arg
485 490 495
Gly Leu Glu Thr Gln Leu Asp Gln Val Leu Lys Glu Arg Arg Ser His
500 505 510
Phe Gly Arg Leu Thr Lys Trp Ala Lys Glu Cys Gly Ile Thr Leu Asp
515 520 525
Pro Leu Gln Pro Leu Ile Glu Ser Glu Lys Gln Arg Val Ala Glu Arg
530 535 540
Gly Ser Ala His Asp Ala Lys Glu Leu Ala Ile Arg Leu Leu Leu Gln
545 550 555 560
Arg Ile Gly Arg Leu Gly His Arg Leu Ser Pro Thr Asn Ala Thr Ala
565 570 575
Ile Gln Glu Leu Leu Arg Pro Val Phe Ala Val Lys Arg Glu Phe Asn
580 585 590
Leu Phe Phe His Asn His Met Gly Ala Leu Tyr Arg Ser Pro Tyr Ser
595 600 605
Thr Ser Arg His Gln Pro Phe Gln Ile Asn Val Asp Val Ala His Gly
610 615 620
Thr Asp Trp Ile Gly Thr Ile Glu Thr Leu Ile Gln Asn Leu Phe Thr
625 630 635 640
Gln Ile Gln Asp Asp Ala Leu Leu Arg Asp Leu Val Gln Leu Glu Gly
645 650 655
Phe Val Phe Ser His Lys Leu Arg Ala Leu Pro Gly Val Ile Pro Ser
660 665 670
Glu Leu Ala Arg Pro Asn Asn Leu Gln Gln Met Gly Leu Pro Ala Leu
675 680 685
Leu Leu Val Leu Leu Gln Ala Asp Gln Val His Arg Glu Thr Val Leu
690 695 700
Arg Val Phe Asn Leu Tyr Gly Ser Ala Ile Asn Gly Tyr Leu Phe Gln
705 710 715 720
Ala Leu Arg Pro Gly Phe Ile Val Arg Ala Gly Phe Gln Arg Leu Glu
725 730 735
Thr Lys Lys Leu Arg Tyr Val Pro Lys Ala Gln Ser Trp Gln Tyr Pro
740 745 750
Asp Arg Leu His His Ala Lys Ser Ala Ile Lys Asn Ser Leu Ser Ala
755 760 765
Gly Trp Ile Lys Lys Asn His Gln Gly Ala Ile Leu Pro Gln Lys Thr
770 775 780
Leu Thr Ala Leu Val Lys Gln Lys Ser Leu Lys Asp Thr Gly Val Pro
785 790 795 800
Glu Tyr Leu Val Gln Ala Pro His Asp Trp Tyr Val Pro Ile Asp Leu
805 810 815
Arg Gly Pro Ala Ile Pro Ile Glu Gly Leu Thr Val Gly Thr Glu Gly
820 825 830
Pro Glu Leu Thr Gln Leu Gly Pro Met Lys Asp Asp Cys Ala Phe Arg
835 840 845
Ala Ile Gly Pro Ser Ser Phe Lys Ser Lys Ile Asp Ala Gly Leu Leu
850 855 860
Pro Gln Asp Val Lys Tyr Gly Asp Met Thr Leu Ile Phe Asp Gln His
865 870 875 880
Tyr Gln Gln Ser Ile Ser Phe Ala Asn Gly Thr Phe Ser Ile Gln Tyr
885 890 895
Gln Pro Thr Ser Leu Gln Val Lys Ala Ala Ile Pro Val Val Asp Lys
900 905 910
Arg Pro Arg Asp Thr Arg Asn Asn Ser His Leu Tyr Asp Arg Ile Val
915 920 925
Ala Ile Asp Leu Gly Glu Arg Lys Ile Gly Tyr Ala Ile Phe Asp Leu
930 935 940
Lys Gln Val Leu Lys Ser Glu Gln Leu Glu Pro Met Arg Glu Asp Gly
945 950 955 960
Lys Pro Leu Ile Gly Ser Ile Ser Ile Arg Ser Ile Arg Gly Leu Met
965 970 975
Lys Ala Val Gln Thr His Arg Asn Arg Arg Gln Pro Asn Tyr Arg Ile
980 985 990
Asp Gln Thr Tyr Ser Lys Ala Leu Met His Tyr Arg Glu Ser Val Ile
995 1000 1005
Gly Asp Val Cys Asn Ala Ile Asp Thr Leu Cys Ala Arg Tyr Gly
1010 1015 1020
Gly Phe Pro Val Leu Glu Ser Ser Val Arg Asn Phe Glu Val Gly
1025 1030 1035
Ser Ala Gln Leu Lys Thr Val Tyr Gly Ser Val Ser Arg Arg Tyr
1040 1045 1050
Thr Trp Ser Ala Val Asp Ala His Lys Asn Gln Arg Gln Gln Tyr
1055 1060 1065
Trp Leu Gly Gly Thr Lys Asp Lys Ile Pro Ile Trp Thr His Pro
1070 1075 1080
Tyr Leu Met Thr Arg Glu Trp Asp Glu Lys Asn Ser Lys Trp Ser
1085 1090 1095
Asn Arg Ser Lys Pro Leu Lys Met His Pro Gly Val Glu Val His
1100 1105 1110
Pro Ala Gly Thr Ser Gln Ile Cys His Gln Cys Lys Arg Asn Pro
1115 1120 1125
Ile Gly Ala Leu Trp Asn Val Ala Asp Thr Val Val Leu Asp Asp
1130 1135 1140
Gln Gly Gln Leu Asp Leu Asp Asp Gly Thr Ile Arg Leu Asn Ser
1145 1150 1155
Gly Tyr Ile Asp Thr Thr Glu Ile Lys Arg Ala Arg Arg Lys Lys
1160 1165 1170
Ile Arg Leu Pro Glu Asn Lys Pro Leu Thr Gly Ser His Lys Thr
1175 1180 1185
Ser His Val Arg Ala Val Ala Arg Arg Asn Leu Arg Gln Pro Pro
1190 1195 1200
Lys Ser Thr Arg Ala Lys Asp Thr Thr Gln Ser Arg Tyr Thr Cys
1205 1210 1215
Leu Tyr Val Asp Cys Gly His Glu Cys His Ala Asp Glu Asn Ala
1220 1225 1230
Ala Ile Asn Ile Gly Arg Lys Tyr Leu Gln Glu Arg Ile His Ile
1235 1240 1245
Glu Ala Ser Arg Gln Ala Leu Ser Thr Arg
1250 1255
<210> 572
<211> 1269
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(1269)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 572
Met Val Ala Gly Leu Lys Lys Ile Lys Arg Asp Gly Val Thr Met Lys
1 5 10 15
Ser Asn Tyr His Gly Gly Val Lys Ala Arg Ala Trp Arg Lys Arg Ile
20 25 30
Gly Gly Leu Ala Arg Arg Gln Lys Glu Thr Val Phe Thr Tyr Lys Phe
35 40 45
Pro Leu Glu Thr Glu Glu Ala Gly Ile Asp Phe Asp Lys Ala Val Gln
50 55 60
Thr Tyr Gly Ile Ala Glu Gly Ile Ser Gln Gly Ser Leu Ile Gly Leu
65 70 75 80
Val Cys Ala Phe His Leu Ser Gly Phe Arg Leu Phe Ser Lys Ala Asp
85 90 95
Glu Thr Lys Ala Phe Cys Asn Gln Gly Arg Tyr Pro Asn Gln Ala Phe
100 105 110
Ala Glu Lys Leu Arg Asn Glu Leu Ser Val Thr Leu Pro Lys Leu Ser
115 120 125
Pro Gln Ser Leu Asp Val Leu Phe Gln Ser Ser Pro Lys Ser Lys Asn
130 135 140
Gly Val Ala Pro Glu Trp Ser Lys Asn Ala Ile Arg Asn Arg Leu Tyr
145 150 155 160
Thr Asn Trp Thr Gly Lys Gly Ala Gly Thr Asn Pro Asp Glu His Leu
165 170 175
Leu Glu Ile Ala Glu Asp Ile Ala Ala Glu Ile Asp Ser Asp Leu Asp
180 185 190
Gly Trp Lys Asp Leu Glu Glu His Pro Glu Lys Gly Leu Ser Ala Ala
195 200 205
Asp Arg Tyr Phe Gln Ala Gln Gly Asp Phe Pro Ser Leu Thr Gly Leu
210 215 220
Pro Pro Ser Val Pro Leu Thr Pro Gln Asn Ser Thr Val Ala Phe Glu
225 230 235 240
Gly Asp Pro Val Cys Leu Asn Pro Ser Asp Asn Thr Leu Leu His Gln
245 250 255
Ala Val Ala Arg Cys Ala Gly Arg Ile Leu Gln Glu Gln Pro Asn Leu
260 265 270
Ser Pro Asp Lys Asn Arg Phe Ile Asn Gln Leu Gln Asp Glu Leu Val
275 280 285
Ser Ser Gln Asn Asn Gly Leu Ser Trp Leu Phe Gly Val Gly Phe Lys
290 295 300
Tyr Trp Lys Glu Met Ser Val Asp Gln Leu Ala Asp Asp Tyr Lys Val
305 310 315 320
Lys Ser Thr Asp Leu Asp Ala Leu Lys Gln Val Lys Ser Phe Ile Asp
325 330 335
Ala Ile Pro Leu Asn Pro Leu Phe Asp Thr Pro His Tyr Gly Glu Phe
340 345 350
Arg Ala Ser Val Ala Gly Lys Met Arg Ser Trp Val Lys Asn Tyr Trp
355 360 365
Lys Arg Leu Leu Asp Leu Lys Ser Gln Leu Gly Thr Ala Asn Ile Asn
370 375 380
Leu Pro Glu Gly Leu Asp Glu Gln Arg Ala Glu Asn Leu Phe Ser Gly
385 390 395 400
Leu Leu Ile Asp Ser Lys Gly Leu Arg Gln Val Thr Asp Lys Leu Pro
405 410 415
Ser Arg Leu Lys Lys Ala Glu Asp Thr Ile Asp Arg Leu Met Gly Asp
420 425 430
Gly Asn Pro Thr Ser Asp Asp Ile Glu Gln Val Glu Thr Val Ala Ala
435 440 445
Glu Ile Ser Ala Phe Ile Gly Gln Val Glu Gln Phe Asn Asn Gln Leu
450 455 460
Glu Gln Arg Leu Glu Asn Pro Leu Glu Gly Asp Asp Glu Thr Phe Leu
465 470 475 480
Lys Gln Leu Lys Ile Asp Leu Pro Ala Glu Phe Lys Lys Pro Pro Ala
485 490 495
Ile Asn Arg Ile Ser Gly Gly Ser Pro Asp Pro Thr Ala Glu Ile Ala
500 505 510
Glu Leu Glu Glu Lys Leu Asp Arg Leu Met Ser Ala Arg Lys Glu His
515 520 525
Tyr Glu Thr Ile Ala Glu Trp Ala Ser Ala Asn Lys Val Thr Leu Asp
530 535 540
Pro Met Glu Ala Met Thr Thr Leu Glu Ala Gln Arg Leu Thr Glu Arg
545 550 555 560
Gly Ala Glu Gly Asp Gln Glu Glu Phe Ala Leu Arg Leu Leu Leu Gln
565 570 575
Arg Ile Gly Arg Leu Ala Asn Arg Leu Ser Pro Gln Gly Ala Thr Ala
580 585 590
Ile Arg Asp Leu Leu Arg Pro Val Phe Thr Glu Lys Arg Glu Phe Asn
595 600 605
Leu Phe Phe His Asn Arg Met Gly Ser Leu Tyr Arg Ser Pro Tyr Ser
610 615 620
Thr Ser Arg His Gln Pro Phe Thr Ile Asp Val Ala Val Ala Lys Asn
625 630 635 640
Thr Asp Trp Met Asp Ala Leu Asp Gly Ile Ala Glu Thr Ile Met Lys
645 650 655
Gly Leu Ser Gln Ala Gly Asp Glu Leu Ser Leu Arg Leu Arg Asp Trp
660 665 670
Ile Asn Ile Ser Gly Phe Ser Leu Ser Gln Arg Leu Arg Gly Leu Pro
675 680 685
Asp Thr Val Pro Gly Glu Leu Ala Leu Val Arg Ser Ala Asp Asp Val
690 695 700
Arg Ile Pro Pro Met Leu Ala Leu Gln Leu Glu Glu Asp Glu Val Ser
705 710 715 720
Arg Glu Val Cys Leu Lys Ala Phe Asn Leu Tyr Val Ser Ala Ile Asn
725 730 735
Gly Cys Leu Phe Arg Ala Leu Arg Glu Gly Phe Ile Val Arg Thr Lys
740 745 750
Phe Gln Arg Leu Glu Arg Asp Val Leu Ser Tyr Val Pro Lys Thr Lys
755 760 765
Leu Trp Asn Tyr Pro Gln Arg Leu Asp Thr Ala Arg Gly Pro Ile His
770 775 780
Ser Ala Leu Ala Ala Ala Trp Ile Asn Lys Glu Gly Ser Val Ile Asp
785 790 795 800
Pro Val Glu Thr Val Thr Ala Leu Ser Asp Thr Gly Phe Ser Asp Asp
805 810 815
Gly Ile Pro Glu Tyr Leu Val Gln Ala Pro His Asp Trp Tyr Thr Pro
820 825 830
Ile Asp Leu Arg Asp Ile Ser Lys Pro Val Ser Gly Leu Pro Val Lys
835 840 845
Lys Asn Ile Thr Gly Leu Lys Arg Gln Lys Lys Gln Thr Ala Phe Arg
850 855 860
Met Val Gly Pro Ser Ser Phe Lys Ser His Leu Asp Ser Thr Leu Leu
865 870 875 880
Ser Glu Glu Val Lys Leu Gly Asp Phe Thr Leu Ile Phe Asp Gln Tyr
885 890 895
Tyr Lys Gln Arg Val Ser Tyr Asn Gly Arg Val Lys Ile Thr Phe Glu
900 905 910
Pro Asp Arg Leu His Val Glu Ala Ala Val Pro Val Ile Asp Lys Arg
915 920 925
Val Arg Pro Ser Thr Glu Glu Asp Ala Leu Phe Asp His Leu Leu Ala
930 935 940
Ile Asp Leu Gly Glu Lys Arg Val Gly Tyr Ala Val Tyr Asp Ile Lys
945 950 955 960
Ala Cys Leu Arg Thr Gly Asp Ile Lys Pro Leu Glu Asp Gly Asp Gly
965 970 975
Lys Pro Ile Val Gly Ser Val Ala Val Pro Ser Ile Arg Arg Leu Met
980 985 990
Lys Ala Val Arg Ser His Arg Gln Gln Arg Gln Pro Asn Gln Lys Val
995 1000 1005
Asn Gln Thr Tyr Ser Thr Ala Leu Met Asn Tyr Arg Glu Asn Val
1010 1015 1020
Ile Gly Asp Val Cys Asn Arg Ile Asp Thr Leu Met Glu Lys Tyr
1025 1030 1035
Asn Ala Phe Pro Val Leu Glu Ser Ser Val Met Asn Phe Glu Ala
1040 1045 1050
Gly Ser Arg Gln Leu Glu Met Val Tyr Gly Ser Val Leu His Arg
1055 1060 1065
Tyr Thr Tyr Ser Lys Ile Asp Ala His Thr Ala Lys Arg Lys Glu
1070 1075 1080
Tyr Trp Tyr Thr Gly Glu Tyr Trp Asp His Pro Tyr Leu Met Ala
1085 1090 1095
His Lys Trp Asn Glu Arg Thr Arg Ser Tyr Ser Gly Ser Leu Ser
1100 1105 1110
Ala Leu Thr Leu Tyr Pro Gly Val Met Val His Pro Ala Gly Thr
1115 1120 1125
Ser Gln Arg Cys His Gln Cys Lys Arg Asn Pro Met Val Glu Ile
1130 1135 1140
Lys Gln Leu Thr Gly Gln Val Glu Ile Asn Ala Asp Gly Ser Leu
1145 1150 1155
Glu Leu Asp Asp Gly Thr Ile Cys Leu Tyr Glu Gly Tyr Asp Tyr
1160 1165 1170
Ser Pro Glu Glu Tyr Lys Lys Ala Lys Arg Glu Lys Arg Arg Leu
1175 1180 1185
Asp Pro Asn Val Pro Leu Ser Gly Arg His Gln Ala Lys His Val
1190 1195 1200
Ser Ala Val Ala Lys Arg Asn Leu Arg Arg Pro Thr Val Ser Met
1205 1210 1215
Met Ser Gly Asp Thr Thr Gln Ala Arg Tyr Val Cys Leu Tyr Thr
1220 1225 1230
Asp Cys Asp Phe Thr Gly His Ala Asp Glu Asn Ala Ala Ile Asn
1235 1240 1245
Ile Gly Trp Lys Tyr Leu Thr Glu Arg Ile Ala Leu Ser Glu Ser
1250 1255 1260
Lys Asp Lys Ala Gly Val
1265
<210> 573
<211> 1285
<212> PRT
<213> Rhodobacter capsulatus
<400> 573
Met Gln Ile Gly Lys Val Gln Gly Arg Thr Ile Ser Glu Phe Gly Asp
1 5 10 15
Pro Ala Gly Gly Leu Lys Arg Lys Ile Ser Thr Asp Gly Lys Asn Arg
20 25 30
Lys Glu Leu Pro Ala His Leu Ser Ser Asp Pro Lys Ala Leu Ile Gly
35 40 45
Gln Trp Ile Ser Gly Ile Asp Lys Ile Tyr Arg Lys Pro Asp Ser Arg
50 55 60
Lys Ser Asp Gly Lys Ala Ile His Ser Pro Thr Pro Ser Lys Met Gln
65 70 75 80
Phe Asp Ala Arg Asp Asp Leu Gly Glu Ala Phe Trp Lys Leu Val Ser
85 90 95
Glu Ala Gly Leu Ala Gln Asp Ser Asp Tyr Asp Gln Phe Lys Arg Arg
100 105 110
Leu His Pro Tyr Gly Asp Lys Phe Gln Pro Ala Asp Ser Gly Ala Lys
115 120 125
Leu Lys Phe Glu Ala Asp Pro Pro Glu Pro Gln Ala Phe His Gly Arg
130 135 140
Trp Tyr Gly Ala Met Ser Lys Arg Gly Asn Asp Ala Lys Glu Leu Ala
145 150 155 160
Ala Ala Leu Tyr Glu His Leu His Val Asp Glu Lys Arg Ile Asp Gly
165 170 175
Gln Pro Lys Arg Asn Pro Lys Thr Asp Lys Phe Ala Pro Gly Leu Val
180 185 190
Val Ala Arg Ala Leu Gly Ile Glu Ser Ser Val Leu Pro Arg Gly Met
195 200 205
Ala Arg Leu Ala Arg Asn Trp Gly Glu Glu Glu Ile Gln Thr Tyr Phe
210 215 220
Val Val Asp Val Ala Ala Ser Val Lys Glu Val Ala Lys Ala Ala Val
225 230 235 240
Ser Ala Ala Gln Ala Phe Asp Pro Pro Arg Gln Val Ser Gly Arg Ser
245 250 255
Leu Ser Pro Lys Val Gly Phe Ala Leu Ala Glu His Leu Glu Arg Val
260 265 270
Thr Gly Ser Lys Arg Cys Ser Phe Asp Pro Ala Ala Gly Pro Ser Val
275 280 285
Leu Ala Leu His Asp Glu Val Lys Lys Thr Tyr Lys Arg Leu Cys Ala
290 295 300
Arg Gly Lys Asn Ala Ala Arg Ala Phe Pro Ala Asp Lys Thr Glu Leu
305 310 315 320
Leu Ala Leu Met Arg His Thr His Glu Asn Arg Val Arg Asn Gln Met
325 330 335
Val Arg Met Gly Arg Val Ser Glu Tyr Arg Gly Gln Gln Ala Gly Asp
340 345 350
Leu Ala Gln Ser His Tyr Trp Thr Ser Ala Gly Gln Thr Glu Ile Lys
355 360 365
Glu Ser Glu Ile Phe Val Arg Leu Trp Val Gly Ala Phe Ala Leu Ala
370 375 380
Gly Arg Ser Met Lys Ala Trp Ile Asp Pro Met Gly Lys Ile Val Asn
385 390 395 400
Thr Glu Lys Asn Asp Arg Asp Leu Thr Ala Ala Val Asn Ile Arg Gln
405 410 415
Val Ile Ser Asn Lys Glu Met Val Ala Glu Ala Met Ala Arg Arg Gly
420 425 430
Ile Tyr Phe Gly Glu Thr Pro Glu Leu Asp Arg Leu Gly Ala Glu Gly
435 440 445
Asn Glu Gly Phe Val Phe Ala Leu Leu Arg Tyr Leu Arg Gly Cys Arg
450 455 460
Asn Gln Thr Phe His Leu Gly Ala Arg Ala Gly Phe Leu Lys Glu Ile
465 470 475 480
Arg Lys Glu Leu Glu Lys Thr Arg Trp Gly Lys Ala Lys Glu Ala Glu
485 490 495
His Val Val Leu Thr Asp Lys Thr Val Ala Ala Ile Arg Ala Ile Ile
500 505 510
Asp Asn Asp Ala Lys Ala Leu Gly Ala Arg Leu Leu Ala Asp Leu Ser
515 520 525
Gly Ala Phe Val Ala His Tyr Ala Ser Lys Glu His Phe Ser Thr Leu
530 535 540
Tyr Ser Glu Ile Val Lys Ala Val Lys Asp Ala Pro Glu Val Ser Ser
545 550 555 560
Gly Leu Pro Arg Leu Lys Leu Leu Leu Lys Arg Ala Asp Gly Val Arg
565 570 575
Gly Tyr Val His Gly Leu Arg Asp Thr Arg Lys His Ala Phe Ala Thr
580 585 590
Lys Leu Pro Pro Pro Pro Ala Pro Arg Glu Leu Asp Asp Pro Ala Thr
595 600 605
Lys Ala Arg Tyr Ile Ala Leu Leu Arg Leu Tyr Asp Gly Pro Phe Arg
610 615 620
Ala Tyr Ala Ser Gly Ile Thr Gly Thr Ala Leu Ala Gly Pro Ala Ala
625 630 635 640
Arg Ala Lys Glu Ala Ala Thr Ala Leu Ala Gln Ser Val Asn Val Thr
645 650 655
Lys Ala Tyr Ser Asp Val Met Glu Gly Arg Ser Ser Arg Leu Arg Pro
660 665 670
Pro Asn Asp Gly Glu Thr Leu Arg Glu Tyr Leu Ser Ala Leu Thr Gly
675 680 685
Glu Thr Ala Thr Glu Phe Arg Val Gln Ile Gly Tyr Glu Ser Asp Ser
690 695 700
Glu Asn Ala Arg Lys Gln Ala Glu Phe Ile Glu Asn Tyr Arg Arg Asp
705 710 715 720
Met Leu Ala Phe Met Phe Glu Asp Tyr Ile Arg Ala Lys Gly Phe Asp
725 730 735
Trp Ile Leu Lys Ile Glu Pro Gly Ala Thr Ala Met Thr Arg Ala Pro
740 745 750
Val Leu Pro Glu Pro Ile Asp Thr Arg Gly Gln Tyr Glu His Trp Gln
755 760 765
Ala Ala Leu Tyr Leu Val Met His Phe Val Pro Ala Ser Asp Val Ser
770 775 780
Asn Leu Leu His Gln Leu Arg Lys Trp Glu Ala Leu Gln Gly Lys Tyr
785 790 795 800
Glu Leu Val Gln Asp Gly Asp Ala Thr Asp Gln Ala Asp Ala Arg Arg
805 810 815
Glu Ala Leu Asp Leu Val Lys Arg Phe Arg Asp Val Leu Val Leu Phe
820 825 830
Leu Lys Thr Gly Glu Ala Arg Phe Glu Gly Arg Ala Ala Pro Phe Asp
835 840 845
Leu Lys Pro Phe Arg Ala Leu Phe Ala Asn Pro Ala Thr Phe Asp Arg
850 855 860
Leu Phe Met Ala Thr Pro Thr Thr Ala Arg Pro Ala Glu Asp Asp Pro
865 870 875 880
Glu Gly Asp Gly Ala Ser Glu Pro Glu Leu Arg Val Ala Arg Thr Leu
885 890 895
Arg Gly Leu Arg Gln Ile Ala Arg Tyr Asn His Met Ala Val Leu Ser
900 905 910
Asp Leu Phe Ala Lys His Lys Val Arg Asp Glu Glu Val Ala Arg Leu
915 920 925
Ala Glu Ile Glu Asp Glu Thr Gln Glu Lys Ser Gln Ile Val Ala Ala
930 935 940
Gln Glu Leu Arg Thr Asp Leu His Asp Lys Val Met Lys Cys His Pro
945 950 955 960
Lys Thr Ile Ser Pro Glu Glu Arg Gln Ser Tyr Ala Ala Ala Ile Lys
965 970 975
Thr Ile Glu Glu His Arg Phe Leu Val Gly Arg Val Tyr Leu Gly Asp
980 985 990
His Leu Arg Leu His Arg Leu Met Met Asp Val Ile Gly Arg Leu Ile
995 1000 1005
Asp Tyr Ala Gly Ala Tyr Glu Arg Asp Thr Gly Thr Phe Leu Ile
1010 1015 1020
Asn Ala Ser Lys Gln Leu Gly Ala Gly Ala Asp Trp Ala Val Thr
1025 1030 1035
Ile Ala Gly Ala Ala Asn Thr Asp Ala Arg Thr Gln Thr Arg Lys
1040 1045 1050
Asp Leu Ala His Phe Asn Val Leu Asp Arg Ala Asp Gly Thr Pro
1055 1060 1065
Asp Leu Thr Ala Leu Val Asn Arg Ala Arg Glu Met Met Ala Tyr
1070 1075 1080
Asp Arg Lys Arg Lys Asn Ala Val Pro Arg Ser Ile Leu Asp Met
1085 1090 1095
Leu Ala Arg Leu Gly Leu Thr Leu Lys Trp Gln Met Lys Asp His
1100 1105 1110
Leu Leu Gln Asp Ala Thr Ile Thr Gln Ala Ala Ile Lys His Leu
1115 1120 1125
Asp Lys Val Arg Leu Thr Val Gly Gly Pro Ala Ala Val Thr Glu
1130 1135 1140
Ala Arg Phe Ser Gln Asp Tyr Leu Gln Met Val Ala Ala Val Phe
1145 1150 1155
Asn Gly Ser Val Gln Asn Pro Lys Pro Arg Arg Arg Asp Asp Gly
1160 1165 1170
Asp Ala Trp His Lys Pro Pro Lys Pro Ala Thr Ala Gln Ser Gln
1175 1180 1185
Pro Asp Gln Lys Pro Pro Asn Lys Ala Pro Ser Ala Gly Ser Arg
1190 1195 1200
Leu Pro Pro Pro Gln Val Gly Glu Val Tyr Glu Gly Val Val Val
1205 1210 1215
Lys Val Ile Asp Thr Gly Ser Leu Gly Phe Leu Ala Val Glu Gly
1220 1225 1230
Val Ala Gly Asn Ile Gly Leu His Ile Ser Arg Leu Arg Arg Ile
1235 1240 1245
Arg Glu Asp Ala Ile Ile Val Gly Arg Arg Tyr Arg Phe Arg Val
1250 1255 1260
Glu Ile Tyr Val Pro Pro Lys Ser Asn Thr Ser Lys Leu Asn Ala
1265 1270 1275
Ala Asp Leu Val Arg Ile Asp
1280 1285
<210> 574
<211> 1340
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(1340)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 574
Met Gln Ile Ser Lys Val Asn His Lys His Val Ala Val Gly Gln Lys
1 5 10 15
Asp Arg Glu Arg Ile Thr Gly Phe Ile Tyr Asn Asp Pro Val Gly Asp
20 25 30
Glu Lys Ser Leu Glu Asp Val Val Ala Lys Arg Ala Asn Asp Thr Lys
35 40 45
Val Leu Phe Asn Val Phe Asn Thr Lys Asp Leu Tyr Asp Ser Gln Glu
50 55 60
Ser Asp Lys Ser Glu Lys Asp Lys Glu Ile Ile Ser Lys Gly Ala Lys
65 70 75 80
Phe Val Ala Lys Ser Phe Asn Ser Ala Ile Thr Ile Leu Lys Lys Gln
85 90 95
Asn Lys Ile Tyr Ser Thr Leu Thr Ser Gln Gln Val Ile Lys Glu Leu
100 105 110
Lys Asp Lys Phe Gly Gly Ala Arg Ile Tyr Asp Asp Asp Ile Glu Glu
115 120 125
Ala Leu Thr Glu Thr Leu Lys Lys Ser Phe Arg Lys Glu Asn Val Arg
130 135 140
Asn Ser Ile Lys Val Leu Ile Glu Asn Ala Ala Gly Ile Arg Ser Ser
145 150 155 160
Leu Ser Lys Asp Glu Glu Glu Leu Ile Gln Glu Tyr Phe Val Lys Gln
165 170 175
Leu Val Glu Glu Tyr Thr Lys Thr Lys Leu Gln Lys Asn Val Val Lys
180 185 190
Ser Ile Lys Asn Gln Asn Met Val Ile Gln Pro Asp Ser Asp Ser Gln
195 200 205
Val Leu Ser Leu Ser Glu Ser Arg Arg Glu Lys Gln Ser Ser Ala Val
210 215 220
Ser Ser Asp Thr Leu Val Asn Cys Lys Glu Lys Asp Val Leu Lys Ala
225 230 235 240
Phe Leu Thr Asp Tyr Ala Val Leu Asp Glu Asp Glu Arg Asn Ser Leu
245 250 255
Leu Trp Lys Leu Arg Asn Leu Val Asn Leu Tyr Phe Tyr Gly Ser Glu
260 265 270
Ser Ile Arg Asp Tyr Ser Tyr Thr Lys Glu Lys Ser Val Trp Lys Glu
275 280 285
His Asp Glu Gln Lys Ala Asn Lys Thr Leu Phe Ile Asp Glu Ile Cys
290 295 300
His Ile Thr Lys Ile Gly Lys Asn Gly Lys Glu Gln Lys Val Leu Asp
305 310 315 320
Tyr Glu Glu Asn Arg Ser Arg Cys Arg Lys Gln Asn Ile Asn Tyr Tyr
325 330 335
Arg Ser Ala Leu Asn Tyr Ala Lys Asn Asn Thr Ser Gly Ile Phe Glu
340 345 350
Asn Glu Asp Ser Asn His Phe Trp Ile His Leu Ile Glu Asn Glu Val
355 360 365
Glu Arg Leu Tyr Asn Gly Ile Glu Asn Gly Glu Glu Phe Lys Phe Glu
370 375 380
Thr Gly Tyr Ile Ser Glu Lys Val Trp Lys Ala Val Ile Asn His Leu
385 390 395 400
Ser Ile Lys Tyr Ile Ala Leu Gly Lys Ala Val Tyr Asn Tyr Ala Met
405 410 415
Lys Glu Leu Ser Ser Pro Gly Asp Ile Glu Pro Gly Lys Ile Asp Asp
420 425 430
Ser Tyr Ile Asn Gly Ile Thr Ser Phe Asp Tyr Glu Ile Ile Lys Ala
435 440 445
Glu Glu Ser Leu Gln Arg Asp Ile Ser Met Asn Val Val Phe Ala Thr
450 455 460
Asn Tyr Leu Ala Cys Ala Thr Val Asp Thr Asp Lys Asp Phe Leu Leu
465 470 475 480
Phe Ser Lys Glu Asp Ile Arg Ser Cys Thr Lys Lys Asp Gly Asn Leu
485 490 495
Cys Lys Asn Ile Met Gln Phe Trp Gly Gly Tyr Ser Thr Trp Lys Asn
500 505 510
Phe Cys Glu Glu Tyr Leu Lys Asp Asp Lys Asp Ala Leu Glu Leu Leu
515 520 525
Tyr Ser Leu Lys Ser Met Leu Tyr Ser Met Arg Asn Ser Ser Phe His
530 535 540
Phe Ser Thr Glu Asn Val Asp Asn Gly Ser Trp Asp Thr Glu Leu Ile
545 550 555 560
Gly Lys Leu Phe Glu Glu Asp Cys Asn Arg Ala Ala Arg Ile Glu Lys
565 570 575
Glu Lys Phe Tyr Asn Asn Asn Leu His Met Phe Tyr Ser Ser Ser Leu
580 585 590
Leu Glu Lys Val Leu Glu Arg Leu Tyr Ser Ser His His Glu Arg Ala
595 600 605
Ser Gln Val Pro Ser Phe Asn Arg Val Phe Val Arg Lys Asn Phe Pro
610 615 620
Ser Ser Leu Ser Glu Gln Arg Ile Thr Pro Lys Phe Thr Asp Ser Lys
625 630 635 640
Asp Glu Gln Ile Trp Gln Ser Ala Val Tyr Tyr Leu Cys Lys Glu Ile
645 650 655
Tyr Tyr Asn Asp Phe Leu Gln Ser Lys Glu Ala Tyr Lys Leu Phe Arg
660 665 670
Glu Gly Val Lys Asn Leu Asp Lys Asn Asp Ile Asn Asn Gln Lys Ala
675 680 685
Ala Asp Ser Phe Lys Gln Ala Val Val Tyr Tyr Gly Lys Ala Ile Gly
690 695 700
Asn Ala Thr Leu Ser Gln Val Cys Gln Ala Ile Met Thr Glu Tyr Asn
705 710 715 720
Arg Gln Asn Asn Asp Gly Leu Lys Lys Lys Ser Ala Tyr Ala Glu Lys
725 730 735
Gln Asn Ser Asn Lys Tyr Lys His Tyr Pro Leu Phe Leu Lys Gln Val
740 745 750
Leu Gln Ser Ala Phe Trp Glu Tyr Leu Asp Glu Asn Lys Glu Ile Tyr
755 760 765
Gly Phe Ile Ser Ala Gln Ile His Lys Ser Asn Val Glu Ile Lys Ala
770 775 780
Glu Asp Phe Ile Ala Asn Tyr Ser Ser Gln Gln Tyr Lys Lys Leu Val
785 790 795 800
Asp Lys Val Lys Lys Thr Pro Glu Leu Gln Lys Trp Tyr Thr Leu Gly
805 810 815
Arg Leu Ile Asn Pro Arg Gln Ala Asn Gln Phe Leu Gly Ser Ile Arg
820 825 830
Asn Tyr Val Gln Phe Val Lys Asp Ile Gln Arg Arg Ala Lys Glu Asn
835 840 845
Gly Asn Pro Ile Arg Asn Tyr Tyr Glu Val Leu Glu Ser Asp Ser Ile
850 855 860
Ile Lys Ile Leu Glu Met Cys Thr Lys Leu Asn Gly Thr Thr Ser Asn
865 870 875 880
Asp Ile His Asp Tyr Phe Arg Asp Glu Asp Glu Tyr Ala Glu Tyr Ile
885 890 895
Ser Gln Phe Val Asn Phe Gly Asp Val His Ser Gly Ala Ala Leu Asn
900 905 910
Ala Phe Cys Asn Ser Glu Ser Glu Gly Lys Lys Asn Gly Ile Tyr Tyr
915 920 925
Asp Gly Ile Asn Pro Ile Val Asn Arg Asn Trp Val Leu Cys Lys Leu
930 935 940
Tyr Gly Ser Pro Asp Leu Ile Ser Lys Ile Ile Ser Arg Val Asn Glu
945 950 955 960
Asn Met Ile His Asp Phe His Lys Gln Glu Asp Leu Ile Arg Glu Tyr
965 970 975
Gln Ile Lys Gly Ile Cys Ser Asn Lys Lys Glu Gln Gln Asp Leu Arg
980 985 990
Thr Phe Gln Val Leu Lys Asn Arg Val Glu Leu Arg Asp Ile Val Glu
995 1000 1005
Tyr Ser Glu Ile Ile Asn Glu Leu Tyr Gly Gln Leu Ile Lys Trp
1010 1015 1020
Cys Tyr Leu Arg Glu Arg Asp Leu Met Tyr Phe Gln Leu Gly Phe
1025 1030 1035
His Tyr Leu Cys Leu Asn Asn Ala Ser Ser Lys Glu Ala Asp Tyr
1040 1045 1050
Ile Lys Ile Asn Val Asp Asp Arg Asn Ile Ser Gly Ala Ile Leu
1055 1060 1065
Tyr Gln Ile Ala Ala Met Tyr Ile Asn Gly Leu Pro Val Tyr Tyr
1070 1075 1080
Lys Lys Asp Asp Met Tyr Val Ala Leu Lys Ser Gly Lys Lys Ala
1085 1090 1095
Ser Asp Glu Leu Asn Ser Asn Glu Gln Thr Ser Lys Lys Ile Asn
1100 1105 1110
Tyr Phe Leu Lys Tyr Gly Asn Asn Ile Leu Gly Asp Lys Lys Asp
1115 1120 1125
Gln Leu Tyr Leu Ala Gly Leu Glu Leu Phe Glu Asn Val Ala Glu
1130 1135 1140
His Glu Asn Ile Ile Ile Phe Arg Asn Glu Ile Asp His Phe His
1145 1150 1155
Tyr Phe Tyr Asp Arg Asp Arg Ser Met Leu Asp Leu Tyr Ser Glu
1160 1165 1170
Val Phe Asp Arg Phe Phe Thr Tyr Asp Met Lys Leu Arg Lys Asn
1175 1180 1185
Val Val Asn Met Leu Tyr Asn Ile Leu Leu Asp His Asn Ile Val
1190 1195 1200
Ser Ser Phe Val Phe Glu Thr Gly Glu Lys Lys Val Gly Arg Gly
1205 1210 1215
Asp Ser Glu Val Ile Lys Pro Ser Ala Lys Ile Arg Leu Arg Ala
1220 1225 1230
Asn Asn Gly Val Ser Ser Asp Val Phe Thr Tyr Lys Val Gly Ser
1235 1240 1245
Lys Asp Glu Leu Lys Ile Ala Thr Leu Pro Ala Lys Asn Glu Glu
1250 1255 1260
Phe Leu Leu Asn Val Ala Arg Leu Ile Tyr Tyr Pro Asp Met Glu
1265 1270 1275
Ala Val Ser Glu Asn Met Val Arg Glu Gly Val Val Lys Val Glu
1280 1285 1290
Lys Ser Asn Asp Lys Lys Gly Lys Ile Ser Arg Gly Ser Asn Thr
1295 1300 1305
Arg Ser Ser Asn Gln Ser Lys Tyr Asn Asn Lys Ser Lys Asn Arg
1310 1315 1320
Met Asn Tyr Ser Met Gly Ser Ile Phe Glu Lys Met Asp Leu Lys
1325 1330 1335
Phe Asp
1340
<210> 575
<211> 1437
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(1437)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 575
Met Lys Ile Ser Lys Val Arg Glu Glu Asn Arg Gly Ala Lys Leu Thr
1 5 10 15
Val Asn Ala Lys Thr Ala Val Val Ser Glu Asn Arg Ser Gln Glu Gly
20 25 30
Ile Leu Tyr Asn Asp Pro Ser Arg Tyr Gly Lys Ser Arg Lys Asn Asp
35 40 45
Glu Asp Arg Asp Arg Tyr Ile Glu Ser Arg Leu Lys Ser Ser Gly Lys
50 55 60
Leu Tyr Arg Ile Phe Asn Glu Asp Lys Asn Lys Arg Glu Thr Asp Glu
65 70 75 80
Leu Gln Trp Phe Leu Ser Glu Ile Val Lys Lys Ile Asn Arg Arg Asn
85 90 95
Gly Leu Val Leu Ser Asp Met Leu Ser Val Asp Asp Arg Ala Phe Glu
100 105 110
Lys Ala Phe Glu Lys Tyr Ala Glu Leu Ser Tyr Thr Asn Arg Arg Asn
115 120 125
Lys Val Ser Gly Ser Pro Ala Phe Glu Thr Cys Gly Val Asp Ala Ala
130 135 140
Thr Ala Glu Arg Leu Lys Gly Ile Ile Ser Glu Thr Asn Phe Ile Asn
145 150 155 160
Arg Ile Lys Asn Asn Ile Asp Asn Lys Val Ser Glu Asp Ile Ile Asp
165 170 175
Arg Ile Ile Ala Lys Tyr Leu Lys Lys Ser Leu Cys Arg Glu Arg Val
180 185 190
Lys Arg Gly Leu Lys Lys Leu Leu Met Asn Ala Phe Asp Leu Pro Tyr
195 200 205
Ser Asp Pro Asp Ile Asp Val Gln Arg Asp Phe Ile Asp Tyr Val Leu
210 215 220
Glu Asp Phe Tyr His Val Arg Ala Lys Ser Gln Val Ser Arg Ser Ile
225 230 235 240
Lys Asn Met Asn Met Pro Val Gln Pro Glu Gly Asp Gly Lys Phe Ala
245 250 255
Ile Thr Val Ser Lys Gly Gly Thr Glu Ser Gly Asn Lys Arg Ser Ala
260 265 270
Glu Lys Glu Ala Phe Lys Lys Phe Leu Ser Asp Tyr Ala Ser Leu Asp
275 280 285
Glu Arg Val Arg Asp Asp Met Leu Arg Arg Met Arg Arg Leu Val Val
290 295 300
Leu Tyr Phe Tyr Gly Ser Asp Asp Ser Lys Leu Ser Asp Val Asn Glu
305 310 315 320
Lys Phe Asp Val Trp Glu Asp His Ala Ala Arg Arg Val Asp Asn Arg
325 330 335
Glu Phe Ile Lys Leu Pro Leu Glu Asn Lys Leu Ala Asn Gly Lys Thr
340 345 350
Asp Lys Asp Ala Glu Arg Ile Arg Lys Asn Thr Val Lys Glu Leu Tyr
355 360 365
Arg Asn Gln Asn Ile Gly Cys Tyr Arg Gln Ala Val Lys Ala Val Glu
370 375 380
Glu Asp Asn Asn Gly Arg Tyr Phe Asp Asp Lys Met Leu Asn Met Phe
385 390 395 400
Phe Ile His Arg Ile Glu Tyr Gly Val Glu Lys Ile Tyr Ala Asn Leu
405 410 415
Lys Gln Val Thr Glu Phe Lys Ala Arg Thr Gly Tyr Leu Ser Glu Lys
420 425 430
Ile Trp Lys Asp Leu Ile Asn Tyr Ile Ser Ile Lys Tyr Ile Ala Met
435 440 445
Gly Lys Ala Val Tyr Asn Tyr Ala Met Asp Glu Leu Asn Ala Ser Asp
450 455 460
Lys Lys Glu Ile Glu Leu Gly Lys Ile Ser Glu Glu Tyr Leu Ser Gly
465 470 475 480
Ile Ser Ser Phe Asp Tyr Glu Leu Ile Lys Ala Glu Glu Met Leu Gln
485 490 495
Arg Glu Thr Ala Val Tyr Val Ala Phe Ala Ala Arg His Leu Ser Ser
500 505 510
Gln Thr Val Glu Leu Asp Ser Glu Asn Ser Asp Phe Leu Leu Leu Lys
515 520 525
Pro Lys Gly Thr Met Asp Lys Asn Asp Lys Asn Lys Leu Ala Ser Asn
530 535 540
Asn Ile Leu Asn Phe Leu Lys Asp Lys Glu Thr Leu Arg Asp Thr Ile
545 550 555 560
Leu Gln Tyr Phe Gly Gly His Ser Leu Trp Thr Asp Phe Pro Phe Asp
565 570 575
Lys Tyr Leu Ala Gly Gly Lys Asp Asp Val Asp Phe Leu Thr Asp Leu
580 585 590
Lys Asp Val Ile Tyr Ser Met Arg Asn Asp Ser Phe His Tyr Ala Thr
595 600 605
Glu Asn His Asn Asn Gly Lys Trp Asn Lys Glu Leu Ile Ser Ala Met
610 615 620
Phe Glu His Glu Thr Glu Arg Met Thr Val Val Met Lys Asp Lys Phe
625 630 635 640
Tyr Ser Asn Asn Leu Pro Met Phe Tyr Lys Asn Asp Asp Leu Lys Lys
645 650 655
Leu Leu Ile Asp Leu Tyr Lys Asp Asn Val Glu Arg Ala Ser Gln Val
660 665 670
Pro Ser Phe Asn Lys Val Phe Val Arg Lys Asn Phe Pro Ala Leu Val
675 680 685
Arg Asp Lys Asp Asn Leu Gly Ile Glu Leu Asp Leu Lys Ala Asp Ala
690 695 700
Asp Lys Gly Glu Asn Glu Leu Lys Phe Tyr Asn Ala Leu Tyr Tyr Met
705 710 715 720
Phe Lys Glu Ile Tyr Tyr Asn Ala Phe Leu Asn Asp Lys Asn Val Arg
725 730 735
Glu Arg Phe Ile Thr Lys Ala Thr Lys Val Ala Asp Asn Tyr Asp Arg
740 745 750
Asn Lys Glu Arg Asn Leu Lys Asp Arg Ile Lys Ser Ala Gly Ser Asp
755 760 765
Glu Lys Lys Lys Leu Arg Glu Gln Leu Gln Asn Tyr Ile Ala Glu Asn
770 775 780
Asp Phe Gly Gln Arg Ile Lys Asn Ile Val Gln Val Asn Pro Asp Tyr
785 790 795 800
Thr Leu Ala Gln Ile Cys Gln Leu Ile Met Thr Glu Tyr Asn Gln Gln
805 810 815
Asn Asn Gly Cys Met Gln Lys Lys Ser Ala Ala Arg Lys Asp Ile Asn
820 825 830
Lys Asp Ser Tyr Gln His Tyr Lys Met Leu Leu Leu Val Asn Leu Arg
835 840 845
Lys Ala Phe Leu Glu Phe Ile Lys Glu Asn Tyr Ala Phe Val Leu Lys
850 855 860
Pro Tyr Lys His Asp Leu Cys Asp Lys Ala Asp Phe Val Pro Asp Phe
865 870 875 880
Ala Lys Tyr Val Lys Pro Tyr Ala Gly Leu Ile Ser Arg Val Ala Gly
885 890 895
Ser Ser Glu Leu Gln Lys Trp Tyr Ile Val Ser Arg Phe Leu Ser Pro
900 905 910
Ala Gln Ala Asn His Met Leu Gly Phe Leu His Ser Tyr Lys Gln Tyr
915 920 925
Val Trp Asp Ile Tyr Arg Arg Ala Ser Glu Thr Gly Thr Glu Ile Asn
930 935 940
His Ser Ile Ala Glu Asp Lys Ile Ala Gly Val Asp Ile Thr Asp Val
945 950 955 960
Asp Ala Val Ile Asp Leu Ser Val Lys Leu Cys Gly Thr Ile Ser Ser
965 970 975
Glu Ile Ser Asp Tyr Phe Lys Asp Asp Glu Val Tyr Ala Glu Tyr Ile
980 985 990
Ser Ser Tyr Leu Asp Phe Glu Tyr Asp Gly Gly Asn Tyr Lys Asp Ser
995 1000 1005
Leu Asn Arg Phe Cys Asn Ser Asp Ala Val Asn Asp Gln Lys Val
1010 1015 1020
Ala Leu Tyr Tyr Asp Gly Glu His Pro Lys Leu Asn Arg Asn Ile
1025 1030 1035
Ile Leu Ser Lys Leu Tyr Gly Glu Arg Arg Phe Leu Glu Lys Ile
1040 1045 1050
Thr Asp Arg Val Ser Arg Ser Asp Ile Val Glu Tyr Tyr Lys Leu
1055 1060 1065
Lys Lys Glu Thr Ser Gln Tyr Gln Thr Lys Gly Ile Phe Asp Ser
1070 1075 1080
Glu Asp Glu Gln Lys Asn Ile Lys Lys Phe Gln Glu Met Lys Asn
1085 1090 1095
Ile Val Glu Phe Arg Asp Leu Met Asp Tyr Ser Glu Ile Ala Asp
1100 1105 1110
Glu Leu Gln Gly Gln Leu Ile Asn Trp Ile Tyr Leu Arg Glu Arg
1115 1120 1125
Asp Leu Met Asn Phe Gln Leu Gly Tyr His Tyr Ala Cys Leu Asn
1130 1135 1140
Asn Asp Ser Asn Lys Gln Ala Thr Tyr Val Thr Leu Asp Tyr Gln
1145 1150 1155
Gly Lys Lys Asn Arg Lys Ile Asn Gly Ala Ile Leu Tyr Gln Ile
1160 1165 1170
Cys Ala Met Tyr Ile Asn Gly Leu Pro Leu Tyr Tyr Val Asp Lys
1175 1180 1185
Asp Ser Ser Glu Trp Thr Val Ser Asp Gly Lys Glu Ser Thr Gly
1190 1195 1200
Ala Lys Ile Gly Glu Phe Tyr Arg Tyr Ala Lys Ser Phe Glu Asn
1205 1210 1215
Thr Ser Asp Cys Tyr Ala Ser Gly Leu Glu Ile Phe Glu Asn Ile
1220 1225 1230
Ser Glu His Asp Asn Ile Thr Glu Leu Arg Asn Tyr Ile Glu His
1235 1240 1245
Phe Arg Tyr Tyr Ser Ser Phe Asp Arg Ser Phe Leu Gly Ile Tyr
1250 1255 1260
Ser Glu Val Phe Asp Arg Phe Phe Thr Tyr Asp Leu Lys Tyr Arg
1265 1270 1275
Lys Asn Val Pro Thr Ile Leu Tyr Asn Ile Leu Leu Gln His Phe
1280 1285 1290
Val Asn Val Arg Phe Glu Phe Val Ser Gly Lys Lys Met Ile Gly
1295 1300 1305
Ile Asp Lys Lys Asp Arg Lys Ile Ala Lys Glu Lys Glu Cys Ala
1310 1315 1320
Arg Ile Thr Ile Arg Glu Lys Asn Gly Val Tyr Ser Glu Gln Phe
1325 1330 1335
Thr Tyr Lys Leu Lys Asn Gly Thr Val Tyr Val Asp Ala Arg Asp
1340 1345 1350
Lys Arg Tyr Leu Gln Ser Ile Ile Arg Leu Leu Phe Tyr Pro Glu
1355 1360 1365
Lys Val Asn Met Asp Glu Met Ile Glu Val Lys Glu Lys Lys Lys
1370 1375 1380
Pro Ser Asp Asn Asn Thr Gly Lys Gly Tyr Ser Lys Arg Asp Arg
1385 1390 1395
Gln Gln Asp Arg Lys Glu Tyr Asp Lys Tyr Lys Glu Lys Lys Lys
1400 1405 1410
Lys Glu Gly Asn Phe Leu Ser Gly Met Gly Gly Asn Ile Asn Trp
1415 1420 1425
Asp Glu Ile Asn Ala Gln Leu Lys Asn
1430 1435
<210> 576
<211> 1385
<212> PRT
<213> Clostridium aminophilum
<400> 576
Met Lys Phe Ser Lys Val Asp His Thr Arg Ser Ala Val Gly Ile Gln
1 5 10 15
Lys Ala Thr Asp Ser Val His Gly Met Leu Tyr Thr Asp Pro Lys Lys
20 25 30
Gln Glu Val Asn Asp Leu Asp Lys Arg Phe Asp Gln Leu Asn Val Lys
35 40 45
Ala Lys Arg Leu Tyr Asn Val Phe Asn Gln Ser Lys Ala Glu Glu Asp
50 55 60
Asp Asp Glu Lys Arg Phe Gly Lys Val Val Lys Lys Leu Asn Arg Glu
65 70 75 80
Leu Lys Asp Leu Leu Phe His Arg Glu Val Ser Arg Tyr Asn Ser Ile
85 90 95
Gly Asn Ala Lys Tyr Asn Tyr Tyr Gly Ile Lys Ser Asn Pro Glu Glu
100 105 110
Ile Val Ser Asn Leu Gly Met Val Glu Ser Leu Lys Gly Glu Arg Asp
115 120 125
Pro Gln Lys Val Ile Ser Lys Leu Leu Leu Tyr Tyr Leu Arg Lys Gly
130 135 140
Leu Lys Pro Gly Thr Asp Gly Leu Arg Met Ile Leu Glu Ala Ser Cys
145 150 155 160
Gly Leu Arg Lys Leu Ser Gly Asp Glu Lys Glu Leu Lys Val Phe Leu
165 170 175
Gln Thr Leu Asp Glu Asp Phe Glu Lys Lys Thr Phe Lys Lys Asn Leu
180 185 190
Ile Arg Ser Ile Glu Asn Gln Asn Met Ala Val Gln Pro Ser Asn Glu
195 200 205
Gly Asp Pro Ile Ile Gly Ile Thr Gln Gly Arg Phe Asn Ser Gln Lys
210 215 220
Asn Glu Glu Lys Ser Ala Ile Glu Arg Met Met Ser Met Tyr Ala Asp
225 230 235 240
Leu Asn Glu Asp His Arg Glu Asp Val Leu Arg Lys Leu Arg Arg Leu
245 250 255
Asn Val Leu Tyr Phe Asn Val Asp Thr Glu Lys Thr Glu Glu Pro Thr
260 265 270
Leu Pro Gly Glu Val Asp Thr Asn Pro Val Phe Glu Val Trp His Asp
275 280 285
His Glu Lys Gly Lys Glu Asn Asp Arg Gln Phe Ala Thr Phe Ala Lys
290 295 300
Ile Leu Thr Glu Asp Arg Glu Thr Arg Lys Lys Glu Lys Leu Ala Val
305 310 315 320
Lys Glu Ala Leu Asn Asp Leu Lys Ser Ala Ile Arg Asp His Asn Ile
325 330 335
Met Ala Tyr Arg Cys Ser Ile Lys Val Thr Glu Gln Asp Lys Asp Gly
340 345 350
Leu Phe Phe Glu Asp Gln Arg Ile Asn Arg Phe Trp Ile His His Ile
355 360 365
Glu Ser Ala Val Glu Arg Ile Leu Ala Ser Ile Asn Pro Glu Lys Leu
370 375 380
Tyr Lys Leu Arg Ile Gly Tyr Leu Gly Glu Lys Val Trp Lys Asp Leu
385 390 395 400
Leu Asn Tyr Leu Ser Ile Lys Tyr Ile Ala Val Gly Lys Ala Val Phe
405 410 415
His Phe Ala Met Glu Asp Leu Gly Lys Thr Gly Gln Asp Ile Glu Leu
420 425 430
Gly Lys Leu Ser Asn Ser Val Ser Gly Gly Leu Thr Ser Phe Asp Tyr
435 440 445
Glu Gln Ile Arg Ala Asp Glu Thr Leu Gln Arg Gln Leu Ser Val Glu
450 455 460
Val Ala Phe Ala Ala Asn Asn Leu Phe Arg Ala Val Val Gly Gln Thr
465 470 475 480
Gly Lys Lys Ile Glu Gln Ser Lys Ser Glu Glu Asn Glu Glu Asp Phe
485 490 495
Leu Leu Trp Lys Ala Glu Lys Ile Ala Glu Ser Ile Lys Lys Glu Gly
500 505 510
Glu Gly Asn Thr Leu Lys Ser Ile Leu Gln Phe Phe Gly Gly Ala Ser
515 520 525
Ser Trp Asp Leu Asn His Phe Cys Ala Ala Tyr Gly Asn Glu Ser Ser
530 535 540
Ala Leu Gly Tyr Glu Thr Lys Phe Ala Asp Asp Leu Arg Lys Ala Ile
545 550 555 560
Tyr Ser Leu Arg Asn Glu Thr Phe His Phe Thr Thr Leu Asn Lys Gly
565 570 575
Ser Phe Asp Trp Asn Ala Lys Leu Ile Gly Asp Met Phe Ser His Glu
580 585 590
Ala Ala Thr Gly Ile Ala Val Glu Arg Thr Arg Phe Tyr Ser Asn Asn
595 600 605
Leu Pro Met Phe Tyr Arg Glu Ser Asp Leu Lys Arg Ile Met Asp His
610 615 620
Leu Tyr Asn Thr Tyr His Pro Arg Ala Ser Gln Val Pro Ser Phe Asn
625 630 635 640
Ser Val Phe Val Arg Lys Asn Phe Arg Leu Phe Leu Ser Asn Thr Leu
645 650 655
Asn Thr Asn Thr Ser Phe Asp Thr Glu Val Tyr Gln Lys Trp Glu Ser
660 665 670
Gly Val Tyr Tyr Leu Phe Lys Glu Ile Tyr Tyr Asn Ser Phe Leu Pro
675 680 685
Ser Gly Asp Ala His His Leu Phe Phe Glu Gly Leu Arg Arg Ile Arg
690 695 700
Lys Glu Ala Asp Asn Leu Pro Ile Val Gly Lys Glu Ala Lys Lys Arg
705 710 715 720
Asn Ala Val Gln Asp Phe Gly Arg Arg Cys Asp Glu Leu Lys Asn Leu
725 730 735
Ser Leu Ser Ala Ile Cys Gln Met Ile Met Thr Glu Tyr Asn Glu Gln
740 745 750
Asn Asn Gly Asn Arg Lys Val Lys Ser Thr Arg Glu Asp Lys Arg Lys
755 760 765
Pro Asp Ile Phe Gln His Tyr Lys Met Leu Leu Leu Arg Thr Leu Gln
770 775 780
Glu Ala Phe Ala Ile Tyr Ile Arg Arg Glu Glu Phe Lys Phe Ile Phe
785 790 795 800
Asp Leu Pro Lys Thr Leu Tyr Val Met Lys Pro Val Glu Glu Phe Leu
805 810 815
Pro Asn Trp Lys Ser Gly Met Phe Asp Ser Leu Val Glu Arg Val Lys
820 825 830
Gln Ser Pro Asp Leu Gln Arg Trp Tyr Val Leu Cys Lys Phe Leu Asn
835 840 845
Gly Arg Leu Leu Asn Gln Leu Ser Gly Val Ile Arg Ser Tyr Ile Gln
850 855 860
Phe Ala Gly Asp Ile Gln Arg Arg Ala Lys Ala Asn His Asn Arg Leu
865 870 875 880
Tyr Met Asp Asn Thr Gln Arg Val Glu Tyr Tyr Ser Asn Val Leu Glu
885 890 895
Val Val Asp Phe Cys Ile Lys Gly Thr Ser Arg Phe Ser Asn Val Phe
900 905 910
Ser Asp Tyr Phe Arg Asp Glu Asp Ala Tyr Ala Asp Tyr Leu Asp Asn
915 920 925
Tyr Leu Gln Phe Lys Asp Glu Lys Ile Ala Glu Val Ser Ser Phe Ala
930 935 940
Ala Leu Lys Thr Phe Cys Asn Glu Glu Glu Val Lys Ala Gly Ile Tyr
945 950 955 960
Met Asp Gly Glu Asn Pro Val Met Gln Arg Asn Ile Val Met Ala Lys
965 970 975
Leu Phe Gly Pro Asp Glu Val Leu Lys Asn Val Val Pro Lys Val Thr
980 985 990
Arg Glu Glu Ile Glu Glu Tyr Tyr Gln Leu Glu Lys Gln Ile Ala Pro
995 1000 1005
Tyr Arg Gln Asn Gly Tyr Cys Lys Ser Glu Glu Asp Gln Lys Lys
1010 1015 1020
Leu Leu Arg Phe Gln Arg Ile Lys Asn Arg Val Glu Phe Gln Thr
1025 1030 1035
Ile Thr Glu Phe Ser Glu Ile Ile Asn Glu Leu Leu Gly Gln Leu
1040 1045 1050
Ile Ser Trp Ser Phe Leu Arg Glu Arg Asp Leu Leu Tyr Phe Gln
1055 1060 1065
Leu Gly Phe His Tyr Leu Cys Leu His Asn Asp Thr Glu Lys Pro
1070 1075 1080
Ala Glu Tyr Lys Glu Ile Ser Arg Glu Asp Gly Thr Val Ile Arg
1085 1090 1095
Asn Ala Ile Leu His Gln Val Ala Ala Met Tyr Val Gly Gly Leu
1100 1105 1110
Pro Val Tyr Thr Leu Ala Asp Lys Lys Leu Ala Ala Phe Glu Lys
1115 1120 1125
Gly Glu Ala Asp Cys Lys Leu Ser Ile Ser Lys Asp Thr Ala Gly
1130 1135 1140
Ala Gly Lys Lys Ile Lys Asp Phe Phe Arg Tyr Ser Lys Tyr Val
1145 1150 1155
Leu Ile Lys Asp Arg Met Leu Thr Asp Gln Asn Gln Lys Tyr Thr
1160 1165 1170
Ile Tyr Leu Ala Gly Leu Glu Leu Phe Glu Asn Thr Asp Glu His
1175 1180 1185
Asp Asn Ile Thr Asp Val Arg Lys Tyr Val Asp His Phe Lys Tyr
1190 1195 1200
Tyr Ala Thr Ser Asp Glu Asn Ala Met Ser Ile Leu Asp Leu Tyr
1205 1210 1215
Ser Glu Ile His Asp Arg Phe Phe Thr Tyr Asp Met Lys Tyr Gln
1220 1225 1230
Lys Asn Val Ala Asn Met Leu Glu Asn Ile Leu Leu Arg His Phe
1235 1240 1245
Val Leu Ile Arg Pro Glu Phe Phe Thr Gly Ser Lys Lys Val Gly
1250 1255 1260
Glu Gly Lys Lys Ile Thr Cys Lys Ala Arg Ala Gln Ile Glu Ile
1265 1270 1275
Ala Glu Asn Gly Met Arg Ser Glu Asp Phe Thr Tyr Lys Leu Ser
1280 1285 1290
Asp Gly Lys Lys Asn Ile Ser Thr Cys Met Ile Ala Ala Arg Asp
1295 1300 1305
Gln Lys Tyr Leu Asn Thr Val Ala Arg Leu Leu Tyr Tyr Pro His
1310 1315 1320
Glu Ala Lys Lys Ser Ile Val Asp Thr Arg Glu Lys Lys Asn Asn
1325 1330 1335
Lys Lys Thr Asn Arg Gly Asp Gly Thr Phe Asn Lys Gln Lys Gly
1340 1345 1350
Thr Ala Arg Lys Glu Lys Asp Asn Gly Pro Arg Glu Phe Asn Asp
1355 1360 1365
Thr Gly Phe Ser Asn Thr Pro Phe Ala Gly Phe Asp Pro Phe Arg
1370 1375 1380
Asn Ser
1385
<210> 577
<211> 1334
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(1334)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 577
Met Lys Ile Ser Lys Val Asp His Thr Arg Met Ala Val Ala Lys Gly
1 5 10 15
Asn Gln His Arg Arg Asp Glu Ile Ser Gly Ile Leu Tyr Lys Asp Pro
20 25 30
Thr Lys Thr Gly Ser Ile Asp Phe Asp Glu Arg Phe Lys Lys Leu Asn
35 40 45
Cys Ser Ala Lys Ile Leu Tyr His Val Phe Asn Gly Ile Ala Glu Gly
50 55 60
Ser Asn Lys Tyr Lys Asn Ile Val Asp Lys Val Asn Asn Asn Leu Asp
65 70 75 80
Arg Val Leu Phe Thr Gly Lys Ser Tyr Asp Arg Lys Ser Ile Ile Asp
85 90 95
Ile Asp Thr Val Leu Arg Asn Val Glu Lys Ile Asn Ala Phe Asp Arg
100 105 110
Ile Ser Thr Glu Glu Arg Glu Gln Ile Ile Asp Asp Leu Leu Glu Ile
115 120 125
Gln Leu Arg Lys Gly Leu Arg Lys Gly Lys Ala Gly Leu Arg Glu Val
130 135 140
Leu Leu Ile Gly Ala Gly Val Ile Val Arg Thr Asp Lys Lys Gln Glu
145 150 155 160
Ile Ala Asp Phe Leu Glu Ile Leu Asp Glu Asp Phe Asn Lys Thr Asn
165 170 175
Gln Ala Lys Asn Ile Lys Leu Ser Ile Glu Asn Gln Gly Leu Val Val
180 185 190
Ser Pro Val Ser Arg Gly Glu Glu Arg Ile Phe Asp Val Ser Gly Ala
195 200 205
Gln Lys Gly Lys Ser Ser Lys Lys Ala Gln Glu Lys Glu Ala Leu Ser
210 215 220
Ala Phe Leu Leu Asp Tyr Ala Asp Leu Asp Lys Asn Val Arg Phe Glu
225 230 235 240
Tyr Leu Arg Lys Ile Arg Arg Leu Ile Asn Leu Tyr Phe Tyr Val Lys
245 250 255
Asn Asp Asp Val Met Ser Leu Thr Glu Ile Pro Ala Glu Val Asn Leu
260 265 270
Glu Lys Asp Phe Asp Ile Trp Arg Asp His Glu Gln Arg Lys Glu Glu
275 280 285
Asn Gly Asp Phe Val Gly Cys Pro Asp Ile Leu Leu Ala Asp Arg Asp
290 295 300
Val Lys Lys Ser Asn Ser Lys Gln Val Lys Ile Ala Glu Arg Gln Leu
305 310 315 320
Arg Glu Ser Ile Arg Glu Lys Asn Ile Lys Arg Tyr Arg Phe Ser Ile
325 330 335
Lys Thr Ile Glu Lys Asp Asp Gly Thr Tyr Phe Phe Ala Asn Lys Gln
340 345 350
Ile Ser Val Phe Trp Ile His Arg Ile Glu Asn Ala Val Glu Arg Ile
355 360 365
Leu Gly Ser Ile Asn Asp Lys Lys Leu Tyr Arg Leu Arg Leu Gly Tyr
370 375 380
Leu Gly Glu Lys Val Trp Lys Asp Ile Leu Asn Phe Leu Ser Ile Lys
385 390 395 400
Tyr Ile Ala Val Gly Lys Ala Val Phe Asn Phe Ala Met Asp Asp Leu
405 410 415
Gln Glu Lys Asp Arg Asp Ile Glu Pro Gly Lys Ile Ser Glu Asn Ala
420 425 430
Val Asn Gly Leu Thr Ser Phe Asp Tyr Glu Gln Ile Lys Ala Asp Glu
435 440 445
Met Leu Gln Arg Glu Val Ala Val Asn Val Ala Phe Ala Ala Asn Asn
450 455 460
Leu Ala Arg Val Thr Val Asp Ile Pro Gln Asn Gly Glu Lys Glu Asp
465 470 475 480
Ile Leu Leu Trp Asn Lys Ser Asp Ile Lys Lys Tyr Lys Lys Asn Ser
485 490 495
Lys Lys Gly Ile Leu Lys Ser Ile Leu Gln Phe Phe Gly Gly Ala Ser
500 505 510
Thr Trp Asn Met Lys Met Phe Glu Ile Ala Tyr His Asp Gln Pro Gly
515 520 525
Asp Tyr Glu Glu Asn Tyr Leu Tyr Asp Ile Ile Gln Ile Ile Tyr Ser
530 535 540
Leu Arg Asn Lys Ser Phe His Phe Lys Thr Tyr Asp His Gly Asp Lys
545 550 555 560
Asn Trp Asn Arg Glu Leu Ile Gly Lys Met Ile Glu His Asp Ala Glu
565 570 575
Arg Val Ile Ser Val Glu Arg Glu Lys Phe His Ser Asn Asn Leu Pro
580 585 590
Met Phe Tyr Lys Asp Ala Asp Leu Lys Lys Ile Leu Asp Leu Leu Tyr
595 600 605
Ser Asp Tyr Ala Gly Arg Ala Ser Gln Val Pro Ala Phe Asn Thr Val
610 615 620
Leu Val Arg Lys Asn Phe Pro Glu Phe Leu Arg Lys Asp Met Gly Tyr
625 630 635 640
Lys Val His Phe Asn Asn Pro Glu Val Glu Asn Gln Trp His Ser Ala
645 650 655
Val Tyr Tyr Leu Tyr Lys Glu Ile Tyr Tyr Asn Leu Phe Leu Arg Asp
660 665 670
Lys Glu Val Lys Asn Leu Phe Tyr Thr Ser Leu Lys Asn Ile Arg Ser
675 680 685
Glu Val Ser Asp Lys Lys Gln Lys Leu Ala Ser Asp Asp Phe Ala Ser
690 695 700
Arg Cys Glu Glu Ile Glu Asp Arg Ser Leu Pro Glu Ile Cys Gln Ile
705 710 715 720
Ile Met Thr Glu Tyr Asn Ala Gln Asn Phe Gly Asn Arg Lys Val Lys
725 730 735
Ser Gln Arg Val Ile Glu Lys Asn Lys Asp Ile Phe Arg His Tyr Lys
740 745 750
Met Leu Leu Ile Lys Thr Leu Ala Gly Ala Phe Ser Leu Tyr Leu Lys
755 760 765
Gln Glu Arg Phe Ala Phe Ile Gly Lys Ala Thr Pro Ile Pro Tyr Glu
770 775 780
Thr Thr Asp Val Lys Asn Phe Leu Pro Glu Trp Lys Ser Gly Met Tyr
785 790 795 800
Ala Ser Phe Val Glu Glu Ile Lys Asn Asn Leu Asp Leu Gln Glu Trp
805 810 815
Tyr Ile Val Gly Arg Phe Leu Asn Gly Arg Met Leu Asn Gln Leu Ala
820 825 830
Gly Ser Leu Arg Ser Tyr Ile Gln Tyr Ala Glu Asp Ile Glu Arg Arg
835 840 845
Ala Ala Glu Asn Arg Asn Lys Leu Phe Ser Lys Pro Asp Glu Lys Ile
850 855 860
Glu Ala Cys Lys Lys Ala Val Arg Val Leu Asp Leu Cys Ile Lys Ile
865 870 875 880
Ser Thr Arg Ile Ser Ala Glu Phe Thr Asp Tyr Phe Asp Ser Glu Asp
885 890 895
Asp Tyr Ala Asp Tyr Leu Glu Lys Tyr Leu Lys Tyr Gln Asp Asp Ala
900 905 910
Ile Lys Glu Leu Ser Gly Ser Ser Tyr Ala Ala Leu Asp His Phe Cys
915 920 925
Asn Lys Asp Asp Leu Lys Phe Asp Ile Tyr Val Asn Ala Gly Gln Lys
930 935 940
Pro Ile Leu Gln Arg Asn Ile Val Met Ala Lys Leu Phe Gly Pro Asp
945 950 955 960
Asn Ile Leu Ser Glu Val Met Glu Lys Val Thr Glu Ser Ala Ile Arg
965 970 975
Glu Tyr Tyr Asp Tyr Leu Lys Lys Val Ser Gly Tyr Arg Val Arg Gly
980 985 990
Lys Cys Ser Thr Glu Lys Glu Gln Glu Asp Leu Leu Lys Phe Gln Arg
995 1000 1005
Leu Lys Asn Ala Val Glu Phe Arg Asp Val Thr Glu Tyr Ala Glu
1010 1015 1020
Val Ile Asn Glu Leu Leu Gly Gln Leu Ile Ser Trp Ser Tyr Leu
1025 1030 1035
Arg Glu Arg Asp Leu Leu Tyr Phe Gln Leu Gly Phe His Tyr Met
1040 1045 1050
Cys Leu Lys Asn Lys Ser Phe Lys Pro Ala Glu Tyr Val Asp Ile
1055 1060 1065
Arg Arg Asn Asn Gly Thr Ile Ile His Asn Ala Ile Leu Tyr Gln
1070 1075 1080
Ile Val Ser Met Tyr Ile Asn Gly Leu Asp Phe Tyr Ser Cys Asp
1085 1090 1095
Lys Glu Gly Lys Thr Leu Lys Pro Ile Glu Thr Gly Lys Gly Val
1100 1105 1110
Gly Ser Lys Ile Gly Gln Phe Ile Lys Tyr Ser Gln Tyr Leu Tyr
1115 1120 1125
Asn Asp Pro Ser Tyr Lys Leu Glu Ile Tyr Asn Ala Gly Leu Glu
1130 1135 1140
Val Phe Glu Asn Ile Asp Glu His Asp Asn Ile Thr Asp Leu Arg
1145 1150 1155
Lys Tyr Val Asp His Phe Lys Tyr Tyr Ala Tyr Gly Asn Lys Met
1160 1165 1170
Ser Leu Leu Asp Leu Tyr Ser Glu Phe Phe Asp Arg Phe Phe Thr
1175 1180 1185
Tyr Asp Met Lys Tyr Gln Lys Asn Val Val Asn Val Leu Glu Asn
1190 1195 1200
Ile Leu Leu Arg His Phe Val Ile Phe Tyr Pro Lys Phe Gly Ser
1205 1210 1215
Gly Lys Lys Asp Val Gly Ile Arg Asp Cys Lys Lys Glu Arg Ala
1220 1225 1230
Gln Ile Glu Ile Ser Glu Gln Ser Leu Thr Ser Glu Asp Phe Met
1235 1240 1245
Phe Lys Leu Asp Asp Lys Ala Gly Glu Glu Ala Lys Lys Phe Pro
1250 1255 1260
Ala Arg Asp Glu Arg Tyr Leu Gln Thr Ile Ala Lys Leu Leu Tyr
1265 1270 1275
Tyr Pro Asn Glu Ile Glu Asp Met Asn Arg Phe Met Lys Lys Gly
1280 1285 1290
Glu Thr Ile Asn Lys Lys Val Gln Phe Asn Arg Lys Lys Lys Ile
1295 1300 1305
Thr Arg Lys Gln Lys Asn Asn Ser Ser Asn Glu Val Leu Ser Ser
1310 1315 1320
Thr Met Gly Tyr Leu Phe Lys Asn Ile Lys Leu
1325 1330
<210> 578
<211> 1175
<212> PRT
<213> Carnobacterium gallinarum
<400> 578
Met Arg Ile Thr Lys Val Lys Ile Lys Leu Asp Asn Lys Leu Tyr Gln
1 5 10 15
Val Thr Met Gln Lys Glu Glu Lys Tyr Gly Thr Leu Lys Leu Asn Glu
20 25 30
Glu Ser Arg Lys Ser Thr Ala Glu Ile Leu Arg Leu Lys Lys Ala Ser
35 40 45
Phe Asn Lys Ser Phe His Ser Lys Thr Ile Asn Ser Gln Lys Glu Asn
50 55 60
Lys Asn Ala Thr Ile Lys Lys Asn Gly Asp Tyr Ile Ser Gln Ile Phe
65 70 75 80
Glu Lys Leu Val Gly Val Asp Thr Asn Lys Asn Ile Arg Lys Pro Lys
85 90 95
Met Ser Leu Thr Asp Leu Lys Asp Leu Pro Lys Lys Asp Leu Ala Leu
100 105 110
Phe Ile Lys Arg Lys Phe Lys Asn Asp Asp Ile Val Glu Ile Lys Asn
115 120 125
Leu Asp Leu Ile Ser Leu Phe Tyr Asn Ala Leu Gln Lys Val Pro Gly
130 135 140
Glu His Phe Thr Asp Glu Ser Trp Ala Asp Phe Cys Gln Glu Met Met
145 150 155 160
Pro Tyr Arg Glu Tyr Lys Asn Lys Phe Ile Glu Arg Lys Ile Ile Leu
165 170 175
Leu Ala Asn Ser Ile Glu Gln Asn Lys Gly Phe Ser Ile Asn Pro Glu
180 185 190
Thr Phe Ser Lys Arg Lys Arg Val Leu His Gln Trp Ala Ile Glu Val
195 200 205
Gln Glu Arg Gly Asp Phe Ser Ile Leu Asp Glu Lys Leu Ser Lys Leu
210 215 220
Ala Glu Ile Tyr Asn Phe Lys Lys Met Cys Lys Arg Val Gln Asp Glu
225 230 235 240
Leu Asn Asp Leu Glu Lys Ser Met Lys Lys Gly Lys Asn Pro Glu Lys
245 250 255
Glu Lys Glu Ala Tyr Lys Lys Gln Lys Asn Phe Lys Ile Lys Thr Ile
260 265 270
Trp Lys Asp Tyr Pro Tyr Lys Thr His Ile Gly Leu Ile Glu Lys Ile
275 280 285
Lys Glu Asn Glu Glu Leu Asn Gln Phe Asn Ile Glu Ile Gly Lys Tyr
290 295 300
Phe Glu His Tyr Phe Pro Ile Lys Lys Glu Arg Cys Thr Glu Asp Glu
305 310 315 320
Pro Tyr Tyr Leu Asn Ser Glu Thr Ile Ala Thr Thr Val Asn Tyr Gln
325 330 335
Leu Lys Asn Ala Leu Ile Ser Tyr Leu Met Gln Ile Gly Lys Tyr Lys
340 345 350
Gln Phe Gly Leu Glu Asn Gln Val Leu Asp Ser Lys Lys Leu Gln Glu
355 360 365
Ile Gly Ile Tyr Glu Gly Phe Gln Thr Lys Phe Met Asp Ala Cys Val
370 375 380
Phe Ala Thr Ser Ser Leu Lys Asn Ile Ile Glu Pro Met Arg Ser Gly
385 390 395 400
Asp Ile Leu Gly Lys Arg Glu Phe Lys Glu Ala Ile Ala Thr Ser Ser
405 410 415
Phe Val Asn Tyr His His Phe Phe Pro Tyr Phe Pro Phe Glu Leu Lys
420 425 430
Gly Met Lys Asp Arg Glu Ser Glu Leu Ile Pro Phe Gly Glu Gln Thr
435 440 445
Glu Ala Lys Gln Met Gln Asn Ile Trp Ala Leu Arg Gly Ser Val Gln
450 455 460
Gln Ile Arg Asn Glu Ile Phe His Ser Phe Asp Lys Asn Gln Lys Phe
465 470 475 480
Asn Leu Pro Gln Leu Asp Lys Ser Asn Phe Glu Phe Asp Ala Ser Glu
485 490 495
Asn Ser Thr Gly Lys Ser Gln Ser Tyr Ile Glu Thr Asp Tyr Lys Phe
500 505 510
Leu Phe Glu Ala Glu Lys Asn Gln Leu Glu Gln Phe Phe Ile Glu Arg
515 520 525
Ile Lys Ser Ser Gly Ala Leu Glu Tyr Tyr Pro Leu Lys Ser Leu Glu
530 535 540
Lys Leu Phe Ala Lys Lys Glu Met Lys Phe Ser Leu Gly Ser Gln Val
545 550 555 560
Val Ala Phe Ala Pro Ser Tyr Lys Lys Leu Val Lys Lys Gly His Ser
565 570 575
Tyr Gln Thr Ala Thr Glu Gly Thr Ala Asn Tyr Leu Gly Leu Ser Tyr
580 585 590
Tyr Asn Arg Tyr Glu Leu Lys Glu Glu Ser Phe Gln Ala Gln Tyr Tyr
595 600 605
Leu Leu Lys Leu Ile Tyr Gln Tyr Val Phe Leu Pro Asn Phe Ser Gln
610 615 620
Gly Asn Ser Pro Ala Phe Arg Glu Thr Val Lys Ala Ile Leu Arg Ile
625 630 635 640
Asn Lys Asp Glu Ala Arg Lys Lys Met Lys Lys Asn Lys Lys Phe Leu
645 650 655
Arg Lys Tyr Ala Phe Glu Gln Val Arg Glu Met Glu Phe Lys Glu Thr
660 665 670
Pro Asp Gln Tyr Met Ser Tyr Leu Gln Ser Glu Met Arg Glu Glu Lys
675 680 685
Val Arg Lys Ala Glu Lys Asn Asp Lys Gly Phe Glu Lys Asn Ile Thr
690 695 700
Met Asn Phe Glu Lys Leu Leu Met Gln Ile Phe Val Lys Gly Phe Asp
705 710 715 720
Val Phe Leu Thr Thr Phe Ala Gly Lys Glu Leu Leu Leu Ser Ser Glu
725 730 735
Glu Lys Val Ile Lys Glu Thr Glu Ile Ser Leu Ser Lys Lys Ile Asn
740 745 750
Glu Arg Glu Lys Thr Leu Lys Ala Ser Ile Gln Val Glu His Gln Leu
755 760 765
Val Ala Thr Asn Ser Ala Ile Ser Tyr Trp Leu Phe Cys Lys Leu Leu
770 775 780
Asp Ser Arg His Leu Asn Glu Leu Arg Asn Glu Met Ile Lys Phe Lys
785 790 795 800
Gln Ser Arg Ile Lys Phe Asn His Thr Gln His Ala Glu Leu Ile Gln
805 810 815
Asn Leu Leu Pro Ile Val Glu Leu Thr Ile Leu Ser Asn Asp Tyr Asp
820 825 830
Glu Lys Asn Asp Ser Gln Asn Val Asp Val Ser Ala Tyr Phe Glu Asp
835 840 845
Lys Ser Leu Tyr Glu Thr Ala Pro Tyr Val Gln Thr Asp Asp Arg Thr
850 855 860
Arg Val Ser Phe Arg Pro Ile Leu Lys Leu Glu Lys Tyr His Thr Lys
865 870 875 880
Ser Leu Ile Glu Ala Leu Leu Lys Asp Asn Pro Gln Phe Arg Val Ala
885 890 895
Ala Thr Asp Ile Gln Glu Trp Met His Lys Arg Glu Glu Ile Gly Glu
900 905 910
Leu Val Glu Lys Arg Lys Asn Leu His Thr Glu Trp Ala Glu Gly Gln
915 920 925
Gln Thr Leu Gly Ala Glu Lys Arg Glu Glu Tyr Arg Asp Tyr Cys Lys
930 935 940
Lys Ile Asp Arg Phe Asn Trp Lys Ala Asn Lys Val Thr Leu Thr Tyr
945 950 955 960
Leu Ser Gln Leu His Tyr Leu Ile Thr Asp Leu Leu Gly Arg Met Val
965 970 975
Gly Phe Ser Ala Leu Phe Glu Arg Asp Leu Val Tyr Phe Ser Arg Ser
980 985 990
Phe Ser Glu Leu Gly Gly Glu Thr Tyr His Ile Ser Asp Tyr Lys Asn
995 1000 1005
Leu Ser Gly Val Leu Arg Leu Asn Ala Glu Val Lys Pro Ile Lys
1010 1015 1020
Ile Lys Asn Ile Lys Val Ile Asp Asn Glu Glu Asn Pro Tyr Lys
1025 1030 1035
Gly Asn Glu Pro Glu Val Lys Pro Phe Leu Asp Arg Leu His Ala
1040 1045 1050
Tyr Leu Glu Asn Val Ile Gly Ile Lys Ala Val His Gly Lys Ile
1055 1060 1065
Arg Asn Gln Thr Ala His Leu Ser Val Leu Gln Leu Glu Leu Ser
1070 1075 1080
Met Ile Glu Ser Met Asn Asn Leu Arg Asp Leu Met Ala Tyr Asp
1085 1090 1095
Arg Lys Leu Lys Asn Ala Val Thr Lys Ser Met Ile Lys Ile Leu
1100 1105 1110
Asp Lys His Gly Met Ile Leu Lys Leu Lys Ile Asp Glu Asn His
1115 1120 1125
Lys Asn Phe Glu Ile Glu Ser Leu Ile Pro Lys Glu Ile Ile His
1130 1135 1140
Leu Lys Asp Lys Ala Ile Lys Thr Asn Gln Val Ser Glu Glu Tyr
1145 1150 1155
Cys Gln Leu Val Leu Ala Leu Leu Thr Thr Asn Pro Gly Asn Gln
1160 1165 1170
Leu Asn
1175
<210> 579
<211> 1164
<212> PRT
<213> Carnobacterium gallinarum
<400> 579
Met Arg Met Thr Lys Val Lys Ile Asn Gly Ser Pro Val Ser Met Asn
1 5 10 15
Arg Ser Lys Leu Asn Gly His Leu Val Trp Asn Gly Thr Thr Asn Thr
20 25 30
Val Asn Ile Leu Thr Lys Lys Glu Gln Ser Phe Ala Ala Ser Phe Leu
35 40 45
Asn Lys Thr Leu Val Lys Ala Asp Gln Val Lys Gly Tyr Lys Val Leu
50 55 60
Ala Glu Asn Ile Phe Ile Ile Phe Glu Gln Leu Glu Lys Ser Asn Ser
65 70 75 80
Glu Lys Pro Ser Val Tyr Leu Asn Asn Ile Arg Arg Leu Lys Glu Ala
85 90 95
Gly Leu Lys Arg Phe Phe Lys Ser Lys Tyr His Glu Glu Ile Lys Tyr
100 105 110
Thr Ser Glu Lys Asn Gln Ser Val Pro Thr Lys Leu Asn Leu Ile Pro
115 120 125
Leu Phe Phe Asn Ala Val Asp Arg Ile Gln Glu Asp Lys Phe Asp Glu
130 135 140
Lys Asn Trp Ser Tyr Phe Cys Lys Glu Met Ser Pro Tyr Leu Asp Tyr
145 150 155 160
Lys Lys Ser Tyr Leu Asn Arg Lys Lys Glu Ile Leu Ala Asn Ser Ile
165 170 175
Gln Gln Asn Arg Gly Phe Ser Met Pro Thr Ala Glu Glu Pro Asn Leu
180 185 190
Leu Ser Lys Arg Lys Gln Leu Phe Gln Gln Trp Ala Met Lys Phe Gln
195 200 205
Glu Ser Pro Leu Ile Gln Gln Asn Asn Phe Ala Val Glu Gln Phe Asn
210 215 220
Lys Glu Phe Ala Asn Lys Ile Asn Glu Leu Ala Ala Val Tyr Asn Val
225 230 235 240
Asp Glu Leu Cys Thr Ala Ile Thr Glu Lys Leu Met Asn Phe Asp Lys
245 250 255
Asp Lys Ser Asn Lys Thr Arg Asn Phe Glu Ile Lys Lys Leu Trp Lys
260 265 270
Gln His Pro His Asn Lys Asp Lys Ala Leu Ile Lys Leu Phe Asn Gln
275 280 285
Glu Gly Asn Glu Ala Leu Asn Gln Phe Asn Ile Glu Leu Gly Lys Tyr
290 295 300
Phe Glu His Tyr Phe Pro Lys Thr Gly Lys Lys Glu Ser Ala Glu Ser
305 310 315 320
Tyr Tyr Leu Asn Pro Gln Thr Ile Ile Lys Thr Val Gly Tyr Gln Leu
325 330 335
Arg Asn Ala Phe Val Gln Tyr Leu Leu Gln Val Gly Lys Leu His Gln
340 345 350
Tyr Asn Lys Gly Val Leu Asp Ser Gln Thr Leu Gln Glu Ile Gly Met
355 360 365
Tyr Glu Gly Phe Gln Thr Lys Phe Met Asp Ala Cys Val Phe Ala Ser
370 375 380
Ser Ser Leu Arg Asn Ile Ile Gln Ala Thr Thr Asn Glu Asp Ile Leu
385 390 395 400
Thr Arg Glu Lys Phe Lys Lys Glu Leu Glu Lys Asn Val Glu Leu Lys
405 410 415
His Asp Leu Phe Phe Lys Thr Glu Ile Val Glu Glu Arg Asp Glu Asn
420 425 430
Pro Ala Lys Lys Ile Ala Met Thr Pro Asn Glu Leu Asp Leu Trp Ala
435 440 445
Ile Arg Gly Ala Val Gln Arg Val Arg Asn Gln Ile Phe His Gln Gln
450 455 460
Ile Asn Lys Arg His Glu Pro Asn Gln Leu Lys Val Gly Ser Phe Glu
465 470 475 480
Asn Gly Asp Leu Gly Asn Val Ser Tyr Gln Lys Thr Ile Tyr Gln Lys
485 490 495
Leu Phe Asp Ala Glu Ile Lys Asp Ile Glu Ile Tyr Phe Ala Glu Lys
500 505 510
Ile Lys Ser Ser Gly Ala Leu Glu Gln Tyr Ser Met Lys Asp Leu Glu
515 520 525
Lys Leu Phe Ser Asn Lys Glu Leu Thr Leu Ser Leu Gly Gly Gln Val
530 535 540
Val Ala Phe Ala Pro Ser Tyr Lys Lys Leu Tyr Lys Gln Gly Tyr Phe
545 550 555 560
Tyr Gln Asn Glu Lys Thr Ile Glu Leu Glu Gln Phe Thr Asp Tyr Asp
565 570 575
Phe Ser Asn Asp Val Phe Lys Ala Asn Tyr Tyr Leu Ile Lys Leu Ile
580 585 590
Tyr His Tyr Val Phe Leu Pro Gln Phe Ser Gln Ala Asn Asn Lys Leu
595 600 605
Phe Lys Asp Thr Val His Tyr Val Ile Gln Gln Asn Lys Glu Leu Asn
610 615 620
Thr Thr Glu Lys Asp Lys Lys Asn Asn Lys Lys Ile Arg Lys Tyr Ala
625 630 635 640
Phe Glu Gln Val Lys Leu Met Lys Asn Glu Ser Pro Glu Lys Tyr Met
645 650 655
Gln Tyr Leu Gln Arg Glu Met Gln Glu Glu Arg Thr Ile Lys Glu Ala
660 665 670
Lys Lys Thr Asn Glu Glu Lys Pro Asn Tyr Asn Phe Glu Lys Leu Leu
675 680 685
Ile Gln Ile Phe Ile Lys Gly Phe Asp Thr Phe Leu Arg Asn Phe Asp
690 695 700
Leu Asn Leu Asn Pro Ala Glu Glu Leu Val Gly Thr Val Lys Glu Lys
705 710 715 720
Ala Glu Gly Leu Arg Lys Arg Lys Glu Arg Ile Ala Lys Ile Leu Asn
725 730 735
Val Asp Glu Gln Ile Lys Thr Gly Asp Glu Glu Ile Ala Phe Trp Ile
740 745 750
Phe Ala Lys Leu Leu Asp Ala Arg His Leu Ser Glu Leu Arg Asn Glu
755 760 765
Met Ile Lys Phe Lys Gln Ser Ser Val Lys Lys Gly Leu Ile Lys Asn
770 775 780
Gly Asp Leu Ile Glu Gln Met Gln Pro Ile Leu Glu Leu Cys Ile Leu
785 790 795 800
Ser Asn Asp Ser Glu Ser Met Glu Lys Glu Ser Phe Asp Lys Ile Glu
805 810 815
Val Phe Leu Glu Lys Val Glu Leu Ala Lys Asn Glu Pro Tyr Met Gln
820 825 830
Glu Asp Lys Leu Thr Pro Val Lys Phe Arg Phe Met Lys Gln Leu Glu
835 840 845
Lys Tyr Gln Thr Arg Asn Phe Ile Glu Asn Leu Val Ile Glu Asn Pro
850 855 860
Glu Phe Lys Val Ser Glu Lys Ile Val Leu Asn Trp His Glu Glu Lys
865 870 875 880
Glu Lys Ile Ala Asp Leu Val Asp Lys Arg Thr Lys Leu His Glu Glu
885 890 895
Trp Ala Ser Lys Ala Arg Glu Ile Glu Glu Tyr Asn Glu Lys Ile Lys
900 905 910
Lys Asn Lys Ser Lys Lys Leu Asp Lys Pro Ala Glu Phe Ala Lys Phe
915 920 925
Ala Glu Tyr Lys Ile Ile Cys Glu Ala Ile Glu Asn Phe Asn Arg Leu
930 935 940
Asp His Lys Val Arg Leu Thr Tyr Leu Lys Asn Leu His Tyr Leu Met
945 950 955 960
Ile Asp Leu Met Gly Arg Met Val Gly Phe Ser Val Leu Phe Glu Arg
965 970 975
Asp Phe Val Tyr Met Gly Arg Ser Tyr Ser Ala Leu Lys Lys Gln Ser
980 985 990
Ile Tyr Leu Asn Asp Tyr Asp Thr Phe Ala Asn Ile Arg Asp Trp Glu
995 1000 1005
Val Asn Glu Asn Lys His Leu Phe Gly Thr Ser Ser Ser Asp Leu
1010 1015 1020
Thr Phe Gln Glu Thr Ala Glu Phe Lys Asn Leu Lys Lys Pro Met
1025 1030 1035
Glu Asn Gln Leu Lys Ala Leu Leu Gly Val Thr Asn His Ser Phe
1040 1045 1050
Glu Ile Arg Asn Asn Ile Ala His Leu His Val Leu Arg Asn Asp
1055 1060 1065
Gly Lys Gly Glu Gly Val Ser Leu Leu Ser Cys Met Asn Asp Leu
1070 1075 1080
Arg Lys Leu Met Ser Tyr Asp Arg Lys Leu Lys Asn Ala Val Thr
1085 1090 1095
Lys Ala Ile Ile Lys Ile Leu Asp Lys His Gly Met Ile Leu Lys
1100 1105 1110
Leu Thr Asn Asn Asp His Thr Lys Pro Phe Glu Ile Glu Ser Leu
1115 1120 1125
Lys Pro Lys Lys Ile Ile His Leu Glu Lys Ser Asn His Ser Phe
1130 1135 1140
Pro Met Asp Gln Val Ser Gln Glu Tyr Cys Asp Leu Val Lys Lys
1145 1150 1155
Met Leu Val Phe Thr Asn
1160
<210> 580
<211> 1154
<212> PRT
<213> Paludibacter propionicigenes
<400> 580
Met Arg Val Ser Lys Val Lys Val Lys Asp Gly Gly Lys Asp Lys Met
1 5 10 15
Val Leu Val His Arg Lys Thr Thr Gly Ala Gln Leu Val Tyr Ser Gly
20 25 30
Gln Pro Val Ser Asn Glu Thr Ser Asn Ile Leu Pro Glu Lys Lys Arg
35 40 45
Gln Ser Phe Asp Leu Ser Thr Leu Asn Lys Thr Ile Ile Lys Phe Asp
50 55 60
Thr Ala Lys Lys Gln Lys Leu Asn Val Asp Gln Tyr Lys Ile Val Glu
65 70 75 80
Lys Ile Phe Lys Tyr Pro Lys Gln Glu Leu Pro Lys Gln Ile Lys Ala
85 90 95
Glu Glu Ile Leu Pro Phe Leu Asn His Lys Phe Gln Glu Pro Val Lys
100 105 110
Tyr Trp Lys Asn Gly Lys Glu Glu Ser Phe Asn Leu Thr Leu Leu Ile
115 120 125
Val Glu Ala Val Gln Ala Gln Asp Lys Arg Lys Leu Gln Pro Tyr Tyr
130 135 140
Asp Trp Lys Thr Trp Tyr Ile Gln Thr Lys Ser Asp Leu Leu Lys Lys
145 150 155 160
Ser Ile Glu Asn Asn Arg Ile Asp Leu Thr Glu Asn Leu Ser Lys Arg
165 170 175
Lys Lys Ala Leu Leu Ala Trp Glu Thr Glu Phe Thr Ala Ser Gly Ser
180 185 190
Ile Asp Leu Thr His Tyr His Lys Val Tyr Met Thr Asp Val Leu Cys
195 200 205
Lys Met Leu Gln Asp Val Lys Pro Leu Thr Asp Asp Lys Gly Lys Ile
210 215 220
Asn Thr Asn Ala Tyr His Arg Gly Leu Lys Lys Ala Leu Gln Asn His
225 230 235 240
Gln Pro Ala Ile Phe Gly Thr Arg Glu Val Pro Asn Glu Ala Asn Arg
245 250 255
Ala Asp Asn Gln Leu Ser Ile Tyr His Leu Glu Val Val Lys Tyr Leu
260 265 270
Glu His Tyr Phe Pro Ile Lys Thr Ser Lys Arg Arg Asn Thr Ala Asp
275 280 285
Asp Ile Ala His Tyr Leu Lys Ala Gln Thr Leu Lys Thr Thr Ile Glu
290 295 300
Lys Gln Leu Val Asn Ala Ile Arg Ala Asn Ile Ile Gln Gln Gly Lys
305 310 315 320
Thr Asn His His Glu Leu Lys Ala Asp Thr Thr Ser Asn Asp Leu Ile
325 330 335
Arg Ile Lys Thr Asn Glu Ala Phe Val Leu Asn Leu Thr Gly Thr Cys
340 345 350
Ala Phe Ala Ala Asn Asn Ile Arg Asn Met Val Asp Asn Glu Gln Thr
355 360 365
Asn Asp Ile Leu Gly Lys Gly Asp Phe Ile Lys Ser Leu Leu Lys Asp
370 375 380
Asn Thr Asn Ser Gln Leu Tyr Ser Phe Phe Phe Gly Glu Gly Leu Ser
385 390 395 400
Thr Asn Lys Ala Glu Lys Glu Thr Gln Leu Trp Gly Ile Arg Gly Ala
405 410 415
Val Gln Gln Ile Arg Asn Asn Val Asn His Tyr Lys Lys Asp Ala Leu
420 425 430
Lys Thr Val Phe Asn Ile Ser Asn Phe Glu Asn Pro Thr Ile Thr Asp
435 440 445
Pro Lys Gln Gln Thr Asn Tyr Ala Asp Thr Ile Tyr Lys Ala Arg Phe
450 455 460
Ile Asn Glu Leu Glu Lys Ile Pro Glu Ala Phe Ala Gln Gln Leu Lys
465 470 475 480
Thr Gly Gly Ala Val Ser Tyr Tyr Thr Ile Glu Asn Leu Lys Ser Leu
485 490 495
Leu Thr Thr Phe Gln Phe Ser Leu Cys Arg Ser Thr Ile Pro Phe Ala
500 505 510
Pro Gly Phe Lys Lys Val Phe Asn Gly Gly Ile Asn Tyr Gln Asn Ala
515 520 525
Lys Gln Asp Glu Ser Phe Tyr Glu Leu Met Leu Glu Gln Tyr Leu Arg
530 535 540
Lys Glu Asn Phe Ala Glu Glu Ser Tyr Asn Ala Arg Tyr Phe Met Leu
545 550 555 560
Lys Leu Ile Tyr Asn Asn Leu Phe Leu Pro Gly Phe Thr Thr Asp Arg
565 570 575
Lys Ala Phe Ala Asp Ser Val Gly Phe Val Gln Met Gln Asn Lys Lys
580 585 590
Gln Ala Glu Lys Val Asn Pro Arg Lys Lys Glu Ala Tyr Ala Phe Glu
595 600 605
Ala Val Arg Pro Met Thr Ala Ala Asp Ser Ile Ala Asp Tyr Met Ala
610 615 620
Tyr Val Gln Ser Glu Leu Met Gln Glu Gln Asn Lys Lys Glu Glu Lys
625 630 635 640
Val Ala Glu Glu Thr Arg Ile Asn Phe Glu Lys Phe Val Leu Gln Val
645 650 655
Phe Ile Lys Gly Phe Asp Ser Phe Leu Arg Ala Lys Glu Phe Asp Phe
660 665 670
Val Gln Met Pro Gln Pro Gln Leu Thr Ala Thr Ala Ser Asn Gln Gln
675 680 685
Lys Ala Asp Lys Leu Asn Gln Leu Glu Ala Ser Ile Thr Ala Asp Cys
690 695 700
Lys Leu Thr Pro Gln Tyr Ala Lys Ala Asp Asp Ala Thr His Ile Ala
705 710 715 720
Phe Tyr Val Phe Cys Lys Leu Leu Asp Ala Ala His Leu Ser Asn Leu
725 730 735
Arg Asn Glu Leu Ile Lys Phe Arg Glu Ser Val Asn Glu Phe Lys Phe
740 745 750
His His Leu Leu Glu Ile Ile Glu Ile Cys Leu Leu Ser Ala Asp Val
755 760 765
Val Pro Thr Asp Tyr Arg Asp Leu Tyr Ser Ser Glu Ala Asp Cys Leu
770 775 780
Ala Arg Leu Arg Pro Phe Ile Glu Gln Gly Ala Asp Ile Thr Asn Trp
785 790 795 800
Ser Asp Leu Phe Val Gln Ser Asp Lys His Ser Pro Val Ile His Ala
805 810 815
Asn Ile Glu Leu Ser Val Lys Tyr Gly Thr Thr Lys Leu Leu Glu Gln
820 825 830
Ile Ile Asn Lys Asp Thr Gln Phe Lys Thr Thr Glu Ala Asn Phe Thr
835 840 845
Ala Trp Asn Thr Ala Gln Lys Ser Ile Glu Gln Leu Ile Lys Gln Arg
850 855 860
Glu Asp His His Glu Gln Trp Val Lys Ala Lys Asn Ala Asp Asp Lys
865 870 875 880
Glu Lys Gln Glu Arg Lys Arg Glu Lys Ser Asn Phe Ala Gln Lys Phe
885 890 895
Ile Glu Lys His Gly Asp Asp Tyr Leu Asp Ile Cys Asp Tyr Ile Asn
900 905 910
Thr Tyr Asn Trp Leu Asp Asn Lys Met His Phe Val His Leu Asn Arg
915 920 925
Leu His Gly Leu Thr Ile Glu Leu Leu Gly Arg Met Ala Gly Phe Val
930 935 940
Ala Leu Phe Asp Arg Asp Phe Gln Phe Phe Asp Glu Gln Gln Ile Ala
945 950 955 960
Asp Glu Phe Lys Leu His Gly Phe Val Asn Leu His Ser Ile Asp Lys
965 970 975
Lys Leu Asn Glu Val Pro Thr Lys Lys Ile Lys Glu Ile Tyr Asp Ile
980 985 990
Arg Asn Lys Ile Ile Gln Ile Asn Gly Asn Lys Ile Asn Glu Ser Val
995 1000 1005
Arg Ala Asn Leu Ile Gln Phe Ile Ser Ser Lys Arg Asn Tyr Tyr
1010 1015 1020
Asn Asn Ala Phe Leu His Val Ser Asn Asp Glu Ile Lys Glu Lys
1025 1030 1035
Gln Met Tyr Asp Ile Arg Asn His Ile Ala His Phe Asn Tyr Leu
1040 1045 1050
Thr Lys Asp Ala Ala Asp Phe Ser Leu Ile Asp Leu Ile Asn Glu
1055 1060 1065
Leu Arg Glu Leu Leu His Tyr Asp Arg Lys Leu Lys Asn Ala Val
1070 1075 1080
Ser Lys Ala Phe Ile Asp Leu Phe Asp Lys His Gly Met Ile Leu
1085 1090 1095
Lys Leu Lys Leu Asn Ala Asp His Lys Leu Lys Val Glu Ser Leu
1100 1105 1110
Glu Pro Lys Lys Ile Tyr His Leu Gly Ser Ser Ala Lys Asp Lys
1115 1120 1125
Pro Glu Tyr Gln Tyr Cys Thr Asn Gln Val Met Met Ala Tyr Cys
1130 1135 1140
Asn Met Cys Arg Ser Leu Leu Glu Met Lys Lys
1145 1150
<210> 581
<211> 1120
<212> PRT
<213> Listeria seeligeri
<400> 581
Met Trp Ile Ser Ile Lys Thr Leu Ile His His Leu Gly Val Leu Phe
1 5 10 15
Phe Cys Asp Tyr Met Tyr Asn Arg Arg Glu Lys Lys Ile Ile Glu Val
20 25 30
Lys Thr Met Arg Ile Thr Lys Val Glu Val Asp Arg Lys Lys Val Leu
35 40 45
Ile Ser Arg Asp Lys Asn Gly Gly Lys Leu Val Tyr Glu Asn Glu Met
50 55 60
Gln Asp Asn Thr Glu Gln Ile Met His His Lys Lys Ser Ser Phe Tyr
65 70 75 80
Lys Ser Val Val Asn Lys Thr Ile Cys Arg Pro Glu Gln Lys Gln Met
85 90 95
Lys Lys Leu Val His Gly Leu Leu Gln Glu Asn Ser Gln Glu Lys Ile
100 105 110
Lys Val Ser Asp Val Thr Lys Leu Asn Ile Ser Asn Phe Leu Asn His
115 120 125
Arg Phe Lys Lys Ser Leu Tyr Tyr Phe Pro Glu Asn Ser Pro Asp Lys
130 135 140
Ser Glu Glu Tyr Arg Ile Glu Ile Asn Leu Ser Gln Leu Leu Glu Asp
145 150 155 160
Ser Leu Lys Lys Gln Gln Gly Thr Phe Ile Cys Trp Glu Ser Phe Ser
165 170 175
Lys Asp Met Glu Leu Tyr Ile Asn Trp Ala Glu Asn Tyr Ile Ser Ser
180 185 190
Lys Thr Lys Leu Ile Lys Lys Ser Ile Arg Asn Asn Arg Ile Gln Ser
195 200 205
Thr Glu Ser Arg Ser Gly Gln Leu Met Asp Arg Tyr Met Lys Asp Ile
210 215 220
Leu Asn Lys Asn Lys Pro Phe Asp Ile Gln Ser Val Ser Glu Lys Tyr
225 230 235 240
Gln Leu Glu Lys Leu Thr Ser Ala Leu Lys Ala Thr Phe Lys Glu Ala
245 250 255
Lys Lys Asn Asp Lys Glu Ile Asn Tyr Lys Leu Lys Ser Thr Leu Gln
260 265 270
Asn His Glu Arg Gln Ile Ile Glu Glu Leu Lys Glu Asn Ser Glu Leu
275 280 285
Asn Gln Phe Asn Ile Glu Ile Arg Lys His Leu Glu Thr Tyr Phe Pro
290 295 300
Ile Lys Lys Thr Asn Arg Lys Val Gly Asp Ile Arg Asn Leu Glu Ile
305 310 315 320
Gly Glu Ile Gln Lys Ile Val Asn His Arg Leu Lys Asn Lys Ile Val
325 330 335
Gln Arg Ile Leu Gln Glu Gly Lys Leu Ala Ser Tyr Glu Ile Glu Ser
340 345 350
Thr Val Asn Ser Asn Ser Leu Gln Lys Ile Lys Ile Glu Glu Ala Phe
355 360 365
Ala Leu Lys Phe Ile Asn Ala Cys Leu Phe Ala Ser Asn Asn Leu Arg
370 375 380
Asn Met Val Tyr Pro Val Cys Lys Lys Asp Ile Leu Met Ile Gly Glu
385 390 395 400
Phe Lys Asn Ser Phe Lys Glu Ile Lys His Lys Lys Phe Ile Arg Gln
405 410 415
Trp Ser Gln Phe Phe Ser Gln Glu Ile Thr Val Asp Asp Ile Glu Leu
420 425 430
Ala Ser Trp Gly Leu Arg Gly Ala Ile Ala Pro Ile Arg Asn Glu Ile
435 440 445
Ile His Leu Lys Lys His Ser Trp Lys Lys Phe Phe Asn Asn Pro Thr
450 455 460
Phe Lys Val Lys Lys Ser Lys Ile Ile Asn Gly Lys Thr Lys Asp Val
465 470 475 480
Thr Ser Glu Phe Leu Tyr Lys Glu Thr Leu Phe Lys Asp Tyr Phe Tyr
485 490 495
Ser Glu Leu Asp Ser Val Pro Glu Leu Ile Ile Asn Lys Met Glu Ser
500 505 510
Ser Lys Ile Leu Asp Tyr Tyr Ser Ser Asp Gln Leu Asn Gln Val Phe
515 520 525
Thr Ile Pro Asn Phe Glu Leu Ser Leu Leu Thr Ser Ala Val Pro Phe
530 535 540
Ala Pro Ser Phe Lys Arg Val Tyr Leu Lys Gly Phe Asp Tyr Gln Asn
545 550 555 560
Gln Asp Glu Ala Gln Pro Asp Tyr Asn Leu Lys Leu Asn Ile Tyr Asn
565 570 575
Glu Lys Ala Phe Asn Ser Glu Ala Phe Gln Ala Gln Tyr Ser Leu Phe
580 585 590
Lys Met Val Tyr Tyr Gln Val Phe Leu Pro Gln Phe Thr Thr Asn Asn
595 600 605
Asp Leu Phe Lys Ser Ser Val Asp Phe Ile Leu Thr Leu Asn Lys Glu
610 615 620
Arg Lys Gly Tyr Ala Lys Ala Phe Gln Asp Ile Arg Lys Met Asn Lys
625 630 635 640
Asp Glu Lys Pro Ser Glu Tyr Met Ser Tyr Ile Gln Ser Gln Leu Met
645 650 655
Leu Tyr Gln Lys Lys Gln Glu Glu Lys Glu Lys Ile Asn His Phe Glu
660 665 670
Lys Phe Ile Asn Gln Val Phe Ile Lys Gly Phe Asn Ser Phe Ile Glu
675 680 685
Lys Asn Arg Leu Thr Tyr Ile Cys His Pro Thr Lys Asn Thr Val Pro
690 695 700
Glu Asn Asp Asn Ile Glu Ile Pro Phe His Thr Asp Met Asp Asp Ser
705 710 715 720
Asn Ile Ala Phe Trp Leu Met Cys Lys Leu Leu Asp Ala Lys Gln Leu
725 730 735
Ser Glu Leu Arg Asn Glu Met Ile Lys Phe Ser Cys Ser Leu Gln Ser
740 745 750
Thr Glu Glu Ile Ser Thr Phe Thr Lys Ala Arg Glu Val Ile Gly Leu
755 760 765
Ala Leu Leu Asn Gly Glu Lys Gly Cys Asn Asp Trp Lys Glu Leu Phe
770 775 780
Asp Asp Lys Glu Ala Trp Lys Lys Asn Met Ser Leu Tyr Val Ser Glu
785 790 795 800
Glu Leu Leu Gln Ser Leu Pro Tyr Thr Gln Glu Asp Gly Gln Thr Pro
805 810 815
Val Ile Asn Arg Ser Ile Asp Leu Val Lys Lys Tyr Gly Thr Glu Thr
820 825 830
Ile Leu Glu Lys Leu Phe Ser Ser Ser Asp Asp Tyr Lys Val Ser Ala
835 840 845
Lys Asp Ile Ala Lys Leu His Glu Tyr Asp Val Thr Glu Lys Ile Ala
850 855 860
Gln Gln Glu Ser Leu His Lys Gln Trp Ile Glu Lys Pro Gly Leu Ala
865 870 875 880
Arg Asp Ser Ala Trp Thr Lys Lys Tyr Gln Asn Val Ile Asn Asp Ile
885 890 895
Ser Asn Tyr Gln Trp Ala Lys Thr Lys Val Glu Leu Thr Gln Val Arg
900 905 910
His Leu His Gln Leu Thr Ile Asp Leu Leu Ser Arg Leu Ala Gly Tyr
915 920 925
Met Ser Ile Ala Asp Arg Asp Phe Gln Phe Ser Ser Asn Tyr Ile Leu
930 935 940
Glu Arg Glu Asn Ser Glu Tyr Arg Val Thr Ser Trp Ile Leu Leu Ser
945 950 955 960
Glu Asn Lys Asn Lys Asn Lys Tyr Asn Asp Tyr Glu Leu Tyr Asn Leu
965 970 975
Lys Asn Ala Ser Ile Lys Val Ser Ser Lys Asn Asp Pro Gln Leu Lys
980 985 990
Val Asp Leu Lys Gln Leu Arg Leu Thr Leu Glu Tyr Leu Glu Leu Phe
995 1000 1005
Asp Asn Arg Leu Lys Glu Lys Arg Asn Asn Ile Ser His Phe Asn
1010 1015 1020
Tyr Leu Asn Gly Gln Leu Gly Asn Ser Ile Leu Glu Leu Phe Asp
1025 1030 1035
Asp Ala Arg Asp Val Leu Ser Tyr Asp Arg Lys Leu Lys Asn Ala
1040 1045 1050
Val Ser Lys Ser Leu Lys Glu Ile Leu Ser Ser His Gly Met Glu
1055 1060 1065
Val Thr Phe Lys Pro Leu Tyr Gln Thr Asn His His Leu Lys Ile
1070 1075 1080
Asp Lys Leu Gln Pro Lys Lys Ile His His Leu Gly Glu Lys Ser
1085 1090 1095
Thr Val Ser Ser Asn Gln Val Ser Asn Glu Tyr Cys Gln Leu Val
1100 1105 1110
Arg Thr Leu Leu Thr Met Lys
1115 1120
<210> 582
<211> 970
<212> PRT
<213> Listeria weihenstephanensis
<400> 582
Met Leu Ala Leu Leu His Gln Glu Val Pro Ser Gln Lys Leu His Asn
1 5 10 15
Leu Lys Ser Leu Asn Thr Glu Ser Leu Thr Lys Leu Phe Lys Pro Lys
20 25 30
Phe Gln Asn Met Ile Ser Tyr Pro Pro Ser Lys Gly Ala Glu His Val
35 40 45
Gln Phe Cys Leu Thr Asp Ile Ala Val Pro Ala Ile Arg Asp Leu Asp
50 55 60
Glu Ile Lys Pro Asp Trp Gly Ile Phe Phe Glu Lys Leu Lys Pro Tyr
65 70 75 80
Thr Asp Trp Ala Glu Ser Tyr Ile His Tyr Lys Gln Thr Thr Ile Gln
85 90 95
Lys Ser Ile Glu Gln Asn Lys Ile Gln Ser Pro Asp Ser Pro Arg Lys
100 105 110
Leu Val Leu Gln Lys Tyr Val Thr Ala Phe Leu Asn Gly Glu Pro Leu
115 120 125
Gly Leu Asp Leu Val Ala Lys Lys Tyr Lys Leu Ala Asp Leu Ala Glu
130 135 140
Ser Phe Lys Val Val Asp Leu Asn Glu Asp Lys Ser Ala Asn Tyr Lys
145 150 155 160
Ile Lys Ala Cys Leu Gln Gln His Gln Arg Asn Ile Leu Asp Glu Leu
165 170 175
Lys Glu Asp Pro Glu Leu Asn Gln Tyr Gly Ile Glu Val Lys Lys Tyr
180 185 190
Ile Gln Arg Tyr Phe Pro Ile Lys Arg Ala Pro Asn Arg Ser Lys His
195 200 205
Ala Arg Ala Asp Phe Leu Lys Lys Glu Leu Ile Glu Ser Thr Val Glu
210 215 220
Gln Gln Phe Lys Asn Ala Val Tyr His Tyr Val Leu Glu Gln Gly Lys
225 230 235 240
Met Glu Ala Tyr Glu Leu Thr Asp Pro Lys Thr Lys Asp Leu Gln Asp
245 250 255
Ile Arg Ser Gly Glu Ala Phe Ser Phe Lys Phe Ile Asn Ala Cys Ala
260 265 270
Phe Ala Ser Asn Asn Leu Lys Met Ile Leu Asn Pro Glu Cys Glu Lys
275 280 285
Asp Ile Leu Gly Lys Gly Asp Phe Lys Lys Asn Leu Pro Asn Ser Thr
290 295 300
Thr Gln Ser Asp Val Val Lys Lys Met Ile Pro Phe Phe Ser Asp Glu
305 310 315 320
Ile Gln Asn Val Asn Phe Asp Glu Ala Ile Trp Ala Ile Arg Gly Ser
325 330 335
Ile Gln Gln Ile Arg Asn Glu Val Tyr His Cys Lys Lys His Ser Trp
340 345 350
Lys Ser Ile Leu Lys Ile Lys Gly Phe Glu Phe Glu Pro Asn Asn Met
355 360 365
Lys Tyr Thr Asp Ser Asp Met Gln Lys Leu Met Asp Lys Asp Ile Ala
370 375 380
Lys Ile Pro Asp Phe Ile Glu Glu Lys Leu Lys Ser Ser Gly Ile Ile
385 390 395 400
Arg Phe Tyr Ser His Asp Lys Leu Gln Ser Ile Trp Glu Met Lys Gln
405 410 415
Gly Phe Ser Leu Leu Thr Thr Asn Ala Pro Phe Val Pro Ser Phe Lys
420 425 430
Arg Val Tyr Ala Lys Gly His Asp Tyr Gln Thr Ser Lys Asn Arg Tyr
435 440 445
Tyr Asp Leu Gly Leu Thr Thr Phe Asp Ile Leu Glu Tyr Gly Glu Glu
450 455 460
Asp Phe Arg Ala Arg Tyr Phe Leu Thr Lys Leu Val Tyr Tyr Gln Gln
465 470 475 480
Phe Met Pro Trp Phe Thr Ala Asp Asn Asn Ala Phe Arg Asp Ala Ala
485 490 495
Asn Phe Val Leu Arg Leu Asn Lys Asn Arg Gln Gln Asp Ala Lys Ala
500 505 510
Phe Ile Asn Ile Arg Glu Val Glu Glu Gly Glu Met Pro Arg Asp Tyr
515 520 525
Met Gly Tyr Val Gln Gly Gln Ile Ala Ile His Glu Asp Ser Thr Glu
530 535 540
Asp Thr Pro Asn His Phe Glu Lys Phe Ile Ser Gln Val Phe Ile Lys
545 550 555 560
Gly Phe Asp Ser His Met Arg Ser Ala Asp Leu Lys Phe Ile Lys Asn
565 570 575
Pro Arg Asn Gln Gly Leu Glu Gln Ser Glu Ile Glu Glu Met Ser Phe
580 585 590
Asp Ile Lys Val Glu Pro Ser Phe Leu Lys Asn Lys Asp Asp Tyr Ile
595 600 605
Ala Phe Trp Thr Phe Cys Lys Met Leu Asp Ala Arg His Leu Ser Glu
610 615 620
Leu Arg Asn Glu Met Ile Lys Tyr Asp Gly His Leu Thr Gly Glu Gln
625 630 635 640
Glu Ile Ile Gly Leu Ala Leu Leu Gly Val Asp Ser Arg Glu Asn Asp
645 650 655
Trp Lys Gln Phe Phe Ser Ser Glu Arg Glu Tyr Glu Lys Ile Met Lys
660 665 670
Gly Tyr Val Gly Glu Glu Leu Tyr Gln Arg Glu Pro Tyr Arg Gln Ser
675 680 685
Asp Gly Lys Thr Pro Ile Leu Phe Arg Gly Val Glu Gln Ala Arg Lys
690 695 700
Tyr Gly Thr Glu Thr Val Ile Gln Arg Leu Phe Asp Ala Ser Pro Glu
705 710 715 720
Phe Lys Val Ser Lys Cys Asn Ile Thr Glu Trp Glu Arg Gln Lys Glu
725 730 735
Thr Ile Glu Glu Thr Ile Glu Arg Arg Lys Glu Leu His Asn Glu Trp
740 745 750
Glu Lys Asn Pro Lys Lys Pro Gln Asn Asn Ala Phe Phe Lys Glu Tyr
755 760 765
Lys Glu Cys Cys Asp Ala Ile Asp Ala Tyr Asn Trp His Lys Asn Lys
770 775 780
Thr Thr Leu Val Tyr Val Asn Glu Leu His His Leu Leu Ile Glu Ile
785 790 795 800
Leu Gly Arg Tyr Val Gly Tyr Val Ala Ile Ala Asp Arg Asp Phe Gln
805 810 815
Cys Met Ala Asn Gln Tyr Phe Lys His Ser Gly Ile Thr Glu Arg Val
820 825 830
Glu Tyr Trp Gly Asp Asn Arg Leu Lys Ser Ile Lys Lys Leu Asp Thr
835 840 845
Phe Leu Lys Lys Glu Gly Leu Phe Val Ser Glu Lys Asn Ala Arg Asn
850 855 860
His Ile Ala His Leu Asn Tyr Leu Ser Leu Lys Ser Glu Cys Thr Leu
865 870 875 880
Leu Tyr Leu Ser Glu Arg Leu Arg Glu Ile Phe Lys Tyr Asp Arg Lys
885 890 895
Leu Lys Asn Ala Val Ser Lys Ser Leu Ile Asp Ile Leu Asp Arg His
900 905 910
Gly Met Ser Val Val Phe Ala Asn Leu Lys Glu Asn Lys His Arg Leu
915 920 925
Val Ile Lys Ser Leu Glu Pro Lys Lys Leu Arg His Leu Gly Glu Lys
930 935 940
Lys Ile Asp Asn Gly Tyr Ile Glu Thr Asn Gln Val Ser Glu Glu Tyr
945 950 955 960
Cys Gly Ile Val Lys Arg Leu Leu Glu Ile
965 970
<210> 583
<211> 1051
<212> PRT
<213> Listeria newyorkensis
<400> 583
Met Lys Ile Thr Lys Met Arg Val Asp Gly Arg Thr Ile Val Met Glu
1 5 10 15
Arg Thr Ser Lys Glu Gly Gln Leu Gly Tyr Glu Gly Ile Asp Gly Asn
20 25 30
Lys Thr Thr Glu Ile Ile Phe Asp Lys Lys Lys Glu Ser Phe Tyr Lys
35 40 45
Ser Ile Leu Asn Lys Thr Val Arg Lys Pro Asp Glu Lys Glu Lys Asn
50 55 60
Arg Arg Lys Gln Ala Ile Asn Lys Ala Ile Asn Lys Glu Ile Thr Glu
65 70 75 80
Leu Met Leu Ala Val Leu His Gln Glu Val Pro Ser Gln Lys Leu His
85 90 95
Asn Leu Lys Ser Leu Asn Thr Glu Ser Leu Thr Lys Leu Phe Lys Pro
100 105 110
Lys Phe Gln Asn Met Ile Ser Tyr Pro Pro Ser Lys Gly Ala Glu His
115 120 125
Val Gln Phe Cys Leu Thr Asp Ile Ala Val Pro Ala Ile Arg Asp Leu
130 135 140
Asp Glu Ile Lys Pro Asp Trp Gly Ile Phe Phe Glu Lys Leu Lys Pro
145 150 155 160
Tyr Thr Asp Trp Ala Glu Ser Tyr Ile His Tyr Lys Gln Thr Thr Ile
165 170 175
Gln Lys Ser Ile Glu Gln Asn Lys Ile Gln Ser Pro Asp Ser Pro Arg
180 185 190
Lys Leu Val Leu Gln Lys Tyr Val Thr Ala Phe Leu Asn Gly Glu Pro
195 200 205
Leu Gly Leu Asp Leu Val Ala Lys Lys Tyr Lys Leu Ala Asp Leu Ala
210 215 220
Glu Ser Phe Lys Leu Val Asp Leu Asn Glu Asp Lys Ser Ala Asn Tyr
225 230 235 240
Lys Ile Lys Ala Cys Leu Gln Gln His Gln Arg Asn Ile Leu Asp Glu
245 250 255
Leu Lys Glu Asp Pro Glu Leu Asn Gln Tyr Gly Ile Glu Val Lys Lys
260 265 270
Tyr Ile Gln Arg Tyr Phe Pro Ile Lys Arg Ala Pro Asn Arg Ser Lys
275 280 285
His Ala Arg Ala Asp Phe Leu Lys Lys Glu Leu Ile Glu Ser Thr Val
290 295 300
Glu Gln Gln Phe Lys Asn Ala Val Tyr His Tyr Val Leu Glu Gln Gly
305 310 315 320
Lys Met Glu Ala Tyr Glu Leu Thr Asp Pro Lys Thr Lys Asp Leu Gln
325 330 335
Asp Ile Arg Ser Gly Glu Ala Phe Ser Phe Lys Phe Ile Asn Ala Cys
340 345 350
Ala Phe Ala Ser Asn Asn Leu Lys Met Ile Leu Asn Pro Glu Cys Glu
355 360 365
Lys Asp Ile Leu Gly Lys Gly Asn Phe Lys Lys Asn Leu Pro Asn Ser
370 375 380
Thr Thr Arg Ser Asp Val Val Lys Lys Met Ile Pro Phe Phe Ser Asp
385 390 395 400
Glu Leu Gln Asn Val Asn Phe Asp Glu Ala Ile Trp Ala Ile Arg Gly
405 410 415
Ser Ile Gln Gln Ile Arg Asn Glu Val Tyr His Cys Lys Lys His Ser
420 425 430
Trp Lys Ser Ile Leu Lys Ile Lys Gly Phe Glu Phe Glu Pro Asn Asn
435 440 445
Met Lys Tyr Ala Asp Ser Asp Met Gln Lys Leu Met Asp Lys Asp Ile
450 455 460
Ala Lys Ile Pro Glu Phe Ile Glu Glu Lys Leu Lys Ser Ser Gly Val
465 470 475 480
Val Arg Phe Tyr Arg His Asp Glu Leu Gln Ser Ile Trp Glu Met Lys
485 490 495
Gln Gly Phe Ser Leu Leu Thr Thr Asn Ala Pro Phe Val Pro Ser Phe
500 505 510
Lys Arg Val Tyr Ala Lys Gly His Asp Tyr Gln Thr Ser Lys Asn Arg
515 520 525
Tyr Tyr Asn Leu Asp Leu Thr Thr Phe Asp Ile Leu Glu Tyr Gly Glu
530 535 540
Glu Asp Phe Arg Ala Arg Tyr Phe Leu Thr Lys Leu Val Tyr Tyr Gln
545 550 555 560
Gln Phe Met Pro Trp Phe Thr Ala Asp Asn Asn Ala Phe Arg Asp Ala
565 570 575
Ala Asn Phe Val Leu Arg Leu Asn Lys Asn Arg Gln Gln Asp Ala Lys
580 585 590
Ala Phe Ile Asn Ile Arg Glu Val Glu Glu Gly Glu Met Pro Arg Asp
595 600 605
Tyr Met Gly Tyr Val Gln Gly Gln Ile Ala Ile His Glu Asp Ser Ile
610 615 620
Glu Asp Thr Pro Asn His Phe Glu Lys Phe Ile Ser Gln Val Phe Ile
625 630 635 640
Lys Gly Phe Asp Arg His Met Arg Ser Ala Asn Leu Lys Phe Ile Lys
645 650 655
Asn Pro Arg Asn Gln Gly Leu Glu Gln Ser Glu Ile Glu Glu Met Ser
660 665 670
Phe Asp Ile Lys Val Glu Pro Ser Phe Leu Lys Asn Lys Asp Asp Tyr
675 680 685
Ile Ala Phe Trp Ile Phe Cys Lys Met Leu Asp Ala Arg His Leu Ser
690 695 700
Glu Leu Arg Asn Glu Met Ile Lys Tyr Asp Gly His Leu Thr Gly Glu
705 710 715 720
Gln Glu Ile Ile Gly Leu Ala Leu Leu Gly Val Asp Ser Arg Glu Asn
725 730 735
Asp Trp Lys Gln Phe Phe Ser Ser Glu Arg Glu Tyr Glu Lys Ile Met
740 745 750
Lys Gly Tyr Val Val Glu Glu Leu Tyr Gln Arg Glu Pro Tyr Arg Gln
755 760 765
Ser Asp Gly Lys Thr Pro Ile Leu Phe Arg Gly Val Glu Gln Ala Arg
770 775 780
Lys Tyr Gly Thr Glu Thr Val Ile Gln Arg Leu Phe Asp Ala Asn Pro
785 790 795 800
Glu Phe Lys Val Ser Lys Cys Asn Leu Ala Glu Trp Glu Arg Gln Lys
805 810 815
Glu Thr Ile Glu Glu Thr Ile Lys Arg Arg Lys Glu Leu His Asn Glu
820 825 830
Trp Ala Lys Asn Pro Lys Lys Pro Gln Asn Asn Ala Phe Phe Lys Glu
835 840 845
Tyr Lys Glu Cys Cys Asp Ala Ile Asp Ala Tyr Asn Trp His Lys Asn
850 855 860
Lys Thr Thr Leu Ala Tyr Val Asn Glu Leu His His Leu Leu Ile Glu
865 870 875 880
Ile Leu Gly Arg Tyr Val Gly Tyr Val Ala Ile Ala Asp Arg Asp Phe
885 890 895
Gln Cys Met Ala Asn Gln Tyr Phe Lys His Ser Gly Ile Thr Glu Arg
900 905 910
Val Glu Tyr Trp Gly Asp Asn Arg Leu Lys Ser Ile Lys Lys Leu Asp
915 920 925
Thr Phe Leu Lys Lys Glu Gly Leu Phe Val Ser Glu Lys Asn Ala Arg
930 935 940
Asn His Ile Ala His Leu Asn Tyr Leu Ser Leu Lys Ser Glu Cys Thr
945 950 955 960
Leu Leu Tyr Leu Ser Glu Arg Leu Arg Glu Ile Phe Lys Tyr Asp Arg
965 970 975
Lys Leu Lys Asn Ala Val Ser Lys Ser Leu Ile Asp Ile Leu Asp Arg
980 985 990
His Gly Met Ser Val Val Phe Ala Asn Leu Lys Glu Asn Lys His Arg
995 1000 1005
Leu Val Ile Lys Ser Leu Glu Pro Lys Lys Leu Arg His Leu Gly
1010 1015 1020
Gly Lys Lys Ile Asp Gly Gly Tyr Ile Glu Thr Asn Gln Val Ser
1025 1030 1035
Glu Glu Tyr Cys Gly Ile Val Lys Arg Leu Leu Glu Met
1040 1045 1050
<210> 584
<211> 1182
<212> PRT
<213> Leptotrichia wadei
<400> 584
Met Tyr Met Lys Ile Thr Lys Ile Asp Gly Val Ser His Tyr Lys Lys
1 5 10 15
Gln Asp Lys Gly Ile Leu Lys Lys Lys Trp Lys Asp Leu Asp Glu Arg
20 25 30
Lys Gln Arg Glu Lys Ile Glu Ala Arg Tyr Asn Lys Gln Ile Glu Ser
35 40 45
Lys Ile Tyr Lys Glu Phe Phe Arg Leu Lys Asn Lys Lys Arg Ile Glu
50 55 60
Lys Glu Glu Asp Gln Asn Ile Lys Ser Leu Tyr Phe Phe Ile Lys Glu
65 70 75 80
Leu Tyr Leu Asn Glu Lys Asn Glu Glu Trp Glu Leu Lys Asn Ile Asn
85 90 95
Leu Glu Ile Leu Asp Asp Lys Glu Arg Val Ile Lys Gly Tyr Lys Phe
100 105 110
Lys Glu Asp Val Tyr Phe Phe Lys Glu Gly Tyr Lys Glu Tyr Tyr Leu
115 120 125
Arg Ile Leu Phe Asn Asn Leu Ile Glu Lys Val Gln Asn Glu Asn Arg
130 135 140
Glu Lys Val Arg Lys Asn Lys Glu Phe Leu Asp Leu Lys Glu Ile Phe
145 150 155 160
Lys Lys Tyr Lys Asn Arg Lys Ile Asp Leu Leu Leu Lys Ser Ile Asn
165 170 175
Asn Asn Lys Ile Asn Leu Glu Tyr Lys Lys Glu Asn Val Asn Glu Glu
180 185 190
Ile Tyr Gly Ile Asn Pro Thr Asn Asp Arg Glu Met Thr Phe Tyr Glu
195 200 205
Leu Leu Lys Glu Ile Ile Glu Lys Lys Asp Glu Gln Lys Ser Ile Leu
210 215 220
Glu Glu Lys Leu Asp Asn Phe Asp Ile Thr Asn Phe Leu Glu Asn Ile
225 230 235 240
Glu Lys Ile Phe Asn Glu Glu Thr Glu Ile Asn Ile Ile Lys Gly Lys
245 250 255
Val Leu Asn Glu Leu Arg Glu Tyr Ile Lys Glu Lys Glu Glu Asn Asn
260 265 270
Ser Asp Asn Lys Leu Lys Gln Ile Tyr Asn Leu Glu Leu Lys Lys Tyr
275 280 285
Ile Glu Asn Asn Phe Ser Tyr Lys Lys Gln Lys Ser Lys Ser Lys Asn
290 295 300
Gly Lys Asn Asp Tyr Leu Tyr Leu Asn Phe Leu Lys Lys Ile Met Phe
305 310 315 320
Ile Glu Glu Val Asp Glu Lys Lys Glu Ile Asn Lys Glu Lys Phe Lys
325 330 335
Asn Lys Ile Asn Ser Asn Phe Lys Asn Leu Phe Val Gln His Ile Leu
340 345 350
Asp Tyr Gly Lys Leu Leu Tyr Tyr Lys Glu Asn Asp Glu Tyr Ile Lys
355 360 365
Asn Thr Gly Gln Leu Glu Thr Lys Asp Leu Glu Tyr Ile Lys Thr Lys
370 375 380
Glu Thr Leu Ile Arg Lys Met Ala Val Leu Val Ser Phe Ala Ala Asn
385 390 395 400
Ser Tyr Tyr Asn Leu Phe Gly Arg Val Ser Gly Asp Ile Leu Gly Thr
405 410 415
Glu Val Val Lys Ser Ser Lys Thr Asn Val Ile Lys Val Gly Ser His
420 425 430
Ile Phe Lys Glu Lys Met Leu Asn Tyr Phe Phe Asp Phe Glu Ile Phe
435 440 445
Asp Ala Asn Lys Ile Val Glu Ile Leu Glu Ser Ile Ser Tyr Ser Ile
450 455 460
Tyr Asn Val Arg Asn Gly Val Gly His Phe Asn Lys Leu Ile Leu Gly
465 470 475 480
Lys Tyr Lys Lys Lys Asp Ile Asn Thr Asn Lys Arg Ile Glu Glu Asp
485 490 495
Leu Asn Asn Asn Glu Glu Ile Lys Gly Tyr Phe Ile Lys Lys Arg Gly
500 505 510
Glu Ile Glu Arg Lys Val Lys Glu Lys Phe Leu Ser Asn Asn Leu Gln
515 520 525
Tyr Tyr Tyr Ser Lys Glu Lys Ile Glu Asn Tyr Phe Glu Val Tyr Glu
530 535 540
Phe Glu Ile Leu Lys Arg Lys Ile Pro Phe Ala Pro Asn Phe Lys Arg
545 550 555 560
Ile Ile Lys Lys Gly Glu Asp Leu Phe Asn Asn Lys Asn Asn Lys Lys
565 570 575
Tyr Glu Tyr Phe Lys Asn Phe Asp Lys Asn Ser Ala Glu Glu Lys Lys
580 585 590
Glu Phe Leu Lys Thr Arg Asn Phe Leu Leu Lys Glu Leu Tyr Tyr Asn
595 600 605
Asn Phe Tyr Lys Glu Phe Leu Ser Lys Lys Glu Glu Phe Glu Lys Ile
610 615 620
Val Leu Glu Val Lys Glu Glu Lys Lys Ser Arg Gly Asn Ile Asn Asn
625 630 635 640
Lys Lys Ser Gly Val Ser Phe Gln Ser Ile Asp Asp Tyr Asp Thr Lys
645 650 655
Ile Asn Ile Ser Asp Tyr Ile Ala Ser Ile His Lys Lys Glu Met Glu
660 665 670
Arg Val Glu Lys Tyr Asn Glu Glu Lys Gln Lys Asp Thr Ala Lys Tyr
675 680 685
Ile Arg Asp Phe Val Glu Glu Ile Phe Leu Thr Gly Phe Ile Asn Tyr
690 695 700
Leu Glu Lys Asp Lys Arg Leu His Phe Leu Lys Glu Glu Phe Ser Ile
705 710 715 720
Leu Cys Asn Asn Asn Asn Asn Val Val Asp Phe Asn Ile Asn Ile Asn
725 730 735
Glu Glu Lys Ile Lys Glu Phe Leu Lys Glu Asn Asp Ser Lys Thr Leu
740 745 750
Asn Leu Tyr Leu Phe Phe Asn Met Ile Asp Ser Lys Arg Ile Ser Glu
755 760 765
Phe Arg Asn Glu Leu Val Lys Tyr Lys Gln Phe Thr Lys Lys Arg Leu
770 775 780
Asp Glu Glu Lys Glu Phe Leu Gly Ile Lys Ile Glu Leu Tyr Glu Thr
785 790 795 800
Leu Ile Glu Phe Val Ile Leu Thr Arg Glu Lys Leu Asp Thr Lys Lys
805 810 815
Ser Glu Glu Ile Asp Ala Trp Leu Val Asp Lys Leu Tyr Val Lys Asp
820 825 830
Ser Asn Glu Tyr Lys Glu Tyr Glu Glu Ile Leu Lys Leu Phe Val Asp
835 840 845
Glu Lys Ile Leu Ser Ser Lys Glu Ala Pro Tyr Tyr Ala Thr Asp Asn
850 855 860
Lys Thr Pro Ile Leu Leu Ser Asn Phe Glu Lys Thr Arg Lys Tyr Gly
865 870 875 880
Thr Gln Ser Phe Leu Ser Glu Ile Gln Ser Asn Tyr Lys Tyr Ser Lys
885 890 895
Val Glu Lys Glu Asn Ile Glu Asp Tyr Asn Lys Lys Glu Glu Ile Glu
900 905 910
Gln Lys Lys Lys Ser Asn Ile Glu Lys Leu Gln Asp Leu Lys Val Glu
915 920 925
Leu His Lys Lys Trp Glu Gln Asn Lys Ile Thr Glu Lys Glu Ile Glu
930 935 940
Lys Tyr Asn Asn Thr Thr Arg Lys Ile Asn Glu Tyr Asn Tyr Leu Lys
945 950 955 960
Asn Lys Glu Glu Leu Gln Asn Val Tyr Leu Leu His Glu Met Leu Ser
965 970 975
Asp Leu Leu Ala Arg Asn Val Ala Phe Phe Asn Lys Trp Glu Arg Asp
980 985 990
Phe Lys Phe Ile Val Ile Ala Ile Lys Gln Phe Leu Arg Glu Asn Asp
995 1000 1005
Lys Glu Lys Val Asn Glu Phe Leu Asn Pro Pro Asp Asn Ser Lys
1010 1015 1020
Gly Lys Lys Val Tyr Phe Ser Val Ser Lys Tyr Lys Asn Thr Val
1025 1030 1035
Glu Asn Ile Asp Gly Ile His Lys Asn Phe Met Asn Leu Ile Phe
1040 1045 1050
Leu Asn Asn Lys Phe Met Asn Arg Lys Ile Asp Lys Met Asn Cys
1055 1060 1065
Ala Ile Trp Val Tyr Phe Arg Asn Tyr Ile Ala His Phe Leu His
1070 1075 1080
Leu His Thr Lys Asn Glu Lys Ile Ser Leu Ile Ser Gln Met Asn
1085 1090 1095
Leu Leu Ile Lys Leu Phe Ser Tyr Asp Lys Lys Val Gln Asn His
1100 1105 1110
Ile Leu Lys Ser Thr Lys Thr Leu Leu Glu Lys Tyr Asn Ile Gln
1115 1120 1125
Ile Asn Phe Glu Ile Ser Asn Asp Lys Asn Glu Val Phe Lys Tyr
1130 1135 1140
Lys Ile Lys Asn Arg Leu Tyr Ser Lys Lys Gly Lys Met Leu Gly
1145 1150 1155
Lys Asn Asn Lys Phe Glu Ile Leu Glu Asn Glu Phe Leu Glu Asn
1160 1165 1170
Val Lys Ala Met Leu Glu Tyr Ser Glu
1175 1180
<210> 585
<211> 1180
<212> PRT
<213> Leptotrichia wadei
<400> 585
Met Lys Ile Thr Lys Ile Asp Gly Val Ser His Tyr Lys Lys Gln Asp
1 5 10 15
Lys Gly Ile Leu Lys Lys Lys Trp Lys Asp Leu Asp Glu Arg Lys Gln
20 25 30
Arg Glu Lys Ile Glu Ala Arg Tyr Asn Lys Gln Ile Glu Ser Lys Ile
35 40 45
Tyr Lys Glu Phe Phe Arg Leu Lys Asn Lys Lys Arg Ile Glu Lys Glu
50 55 60
Glu Asp Gln Asn Ile Lys Ser Leu Tyr Phe Phe Ile Lys Glu Leu Tyr
65 70 75 80
Leu Asn Glu Lys Asn Glu Glu Trp Glu Leu Lys Asn Ile Asn Leu Glu
85 90 95
Ile Leu Asp Asp Lys Glu Arg Val Ile Lys Gly Tyr Lys Phe Lys Glu
100 105 110
Asp Val Tyr Phe Phe Lys Glu Gly Tyr Lys Glu Tyr Tyr Leu Arg Ile
115 120 125
Leu Phe Asn Asn Leu Ile Glu Lys Val Gln Asn Glu Asn Arg Glu Lys
130 135 140
Val Arg Lys Asn Lys Glu Phe Leu Asp Leu Lys Glu Ile Phe Lys Lys
145 150 155 160
Tyr Lys Asn Arg Lys Ile Asp Leu Leu Leu Lys Ser Ile Asn Asn Asn
165 170 175
Lys Ile Asn Leu Glu Tyr Lys Lys Glu Asn Val Asn Glu Glu Ile Tyr
180 185 190
Gly Ile Asn Pro Thr Asn Asp Arg Glu Met Thr Phe Tyr Glu Leu Leu
195 200 205
Lys Glu Ile Ile Glu Lys Lys Asp Glu Gln Lys Ser Ile Leu Glu Glu
210 215 220
Lys Leu Asp Asn Phe Asp Ile Thr Asn Phe Leu Glu Asn Ile Glu Lys
225 230 235 240
Ile Phe Asn Glu Glu Thr Glu Ile Asn Ile Ile Lys Gly Lys Val Leu
245 250 255
Asn Glu Leu Arg Glu Tyr Ile Lys Glu Lys Glu Glu Asn Asn Ser Asp
260 265 270
Asn Lys Leu Lys Gln Ile Tyr Asn Leu Glu Leu Lys Lys Tyr Ile Glu
275 280 285
Asn Asn Phe Ser Tyr Lys Lys Gln Lys Ser Lys Ser Lys Asn Gly Lys
290 295 300
Asn Asp Tyr Leu Tyr Leu Asn Phe Leu Lys Lys Ile Met Phe Ile Glu
305 310 315 320
Glu Val Asp Glu Lys Lys Glu Ile Asn Lys Glu Lys Phe Lys Asn Lys
325 330 335
Ile Asn Ser Asn Phe Lys Asn Leu Phe Val Gln His Ile Leu Asp Tyr
340 345 350
Gly Lys Leu Leu Tyr Tyr Lys Glu Asn Asp Glu Tyr Ile Lys Asn Thr
355 360 365
Gly Gln Leu Glu Thr Lys Asp Leu Glu Tyr Ile Lys Thr Lys Glu Thr
370 375 380
Leu Ile Arg Lys Met Ala Val Leu Val Ser Phe Ala Ala Asn Ser Tyr
385 390 395 400
Tyr Asn Leu Phe Gly Arg Val Ser Gly Asp Ile Leu Gly Thr Glu Val
405 410 415
Val Lys Ser Ser Lys Thr Asn Val Ile Lys Val Gly Ser His Ile Phe
420 425 430
Lys Glu Lys Met Leu Asn Tyr Phe Phe Asp Phe Glu Ile Phe Asp Ala
435 440 445
Asn Lys Ile Val Glu Ile Leu Glu Ser Ile Ser Tyr Ser Ile Tyr Asn
450 455 460
Val Arg Asn Gly Val Gly His Phe Asn Lys Leu Ile Leu Gly Lys Tyr
465 470 475 480
Lys Lys Lys Asp Ile Asn Thr Asn Lys Arg Ile Glu Glu Asp Leu Asn
485 490 495
Asn Asn Glu Glu Ile Lys Gly Tyr Phe Ile Lys Lys Arg Gly Glu Ile
500 505 510
Glu Arg Lys Val Lys Glu Lys Phe Leu Ser Asn Asn Leu Gln Tyr Tyr
515 520 525
Tyr Ser Lys Glu Lys Ile Glu Asn Tyr Phe Glu Val Tyr Glu Phe Glu
530 535 540
Ile Leu Lys Arg Lys Ile Pro Phe Ala Pro Asn Phe Lys Arg Ile Ile
545 550 555 560
Lys Lys Gly Glu Asp Leu Phe Asn Asn Lys Asn Asn Lys Lys Tyr Glu
565 570 575
Tyr Phe Lys Asn Phe Asp Lys Asn Ser Ala Glu Glu Lys Lys Glu Phe
580 585 590
Leu Lys Thr Arg Asn Phe Leu Leu Lys Glu Leu Tyr Tyr Asn Asn Phe
595 600 605
Tyr Lys Glu Phe Leu Ser Lys Lys Glu Glu Phe Glu Lys Ile Val Leu
610 615 620
Glu Val Lys Glu Glu Lys Lys Ser Arg Gly Asn Ile Asn Asn Lys Lys
625 630 635 640
Ser Gly Val Ser Phe Gln Ser Ile Asp Asp Tyr Asp Thr Lys Ile Asn
645 650 655
Ile Ser Asp Tyr Ile Ala Ser Ile His Lys Lys Glu Met Glu Arg Val
660 665 670
Glu Lys Tyr Asn Glu Glu Lys Gln Lys Asp Thr Ala Lys Tyr Ile Arg
675 680 685
Asp Phe Val Glu Glu Ile Phe Leu Thr Gly Phe Ile Asn Tyr Leu Glu
690 695 700
Lys Asp Lys Arg Leu His Phe Leu Lys Glu Glu Phe Ser Ile Leu Cys
705 710 715 720
Asn Asn Asn Asn Asn Val Val Asp Phe Asn Ile Asn Ile Asn Glu Glu
725 730 735
Lys Ile Lys Glu Phe Leu Lys Glu Asn Asp Ser Lys Thr Leu Asn Leu
740 745 750
Tyr Leu Phe Phe Asn Met Ile Asp Ser Lys Arg Ile Ser Glu Phe Arg
755 760 765
Asn Glu Leu Val Lys Tyr Lys Gln Phe Thr Lys Lys Arg Leu Asp Glu
770 775 780
Glu Lys Glu Phe Leu Gly Ile Lys Ile Glu Leu Tyr Glu Thr Leu Ile
785 790 795 800
Glu Phe Val Ile Leu Thr Arg Glu Lys Leu Asp Thr Lys Lys Ser Glu
805 810 815
Glu Ile Asp Ala Trp Leu Val Asp Lys Leu Tyr Val Lys Asp Ser Asn
820 825 830
Glu Tyr Lys Glu Tyr Glu Glu Ile Leu Lys Leu Phe Val Asp Glu Lys
835 840 845
Ile Leu Ser Ser Lys Glu Ala Pro Tyr Tyr Ala Thr Asp Asn Lys Thr
850 855 860
Pro Ile Leu Leu Ser Asn Phe Glu Lys Thr Arg Lys Tyr Gly Thr Gln
865 870 875 880
Ser Phe Leu Ser Glu Ile Gln Ser Asn Tyr Lys Tyr Ser Lys Val Glu
885 890 895
Lys Glu Asn Ile Glu Asp Tyr Asn Lys Lys Glu Glu Ile Glu Gln Lys
900 905 910
Lys Lys Ser Asn Ile Glu Lys Leu Gln Asp Leu Lys Val Glu Leu His
915 920 925
Lys Lys Trp Glu Gln Asn Lys Ile Thr Glu Lys Glu Ile Glu Lys Tyr
930 935 940
Asn Asn Thr Thr Arg Lys Ile Asn Glu Tyr Asn Tyr Leu Lys Asn Lys
945 950 955 960
Glu Glu Leu Gln Asn Val Tyr Leu Leu His Glu Met Leu Ser Asp Leu
965 970 975
Leu Ala Arg Asn Val Ala Phe Phe Asn Lys Trp Glu Arg Asp Phe Lys
980 985 990
Phe Ile Val Ile Ala Ile Lys Gln Phe Leu Arg Glu Asn Asp Lys Glu
995 1000 1005
Lys Val Asn Glu Phe Leu Asn Pro Pro Asp Asn Ser Lys Gly Lys
1010 1015 1020
Lys Val Tyr Phe Ser Val Ser Lys Tyr Lys Asn Thr Val Glu Asn
1025 1030 1035
Ile Asp Gly Ile His Lys Asn Phe Met Asn Leu Ile Phe Leu Asn
1040 1045 1050
Asn Lys Phe Met Asn Arg Lys Ile Asp Lys Met Asn Cys Ala Ile
1055 1060 1065
Trp Val Tyr Phe Arg Asn Tyr Ile Ala His Phe Leu His Leu His
1070 1075 1080
Thr Lys Asn Glu Lys Ile Ser Leu Ile Ser Gln Met Asn Leu Leu
1085 1090 1095
Ile Lys Leu Phe Ser Tyr Asp Lys Lys Val Gln Asn His Ile Leu
1100 1105 1110
Lys Ser Thr Lys Thr Leu Leu Glu Lys Tyr Asn Ile Gln Ile Asn
1115 1120 1125
Phe Glu Ile Ser Asn Asp Lys Asn Glu Val Phe Lys Tyr Lys Ile
1130 1135 1140
Lys Asn Arg Leu Tyr Ser Lys Lys Gly Lys Met Leu Gly Lys Asn
1145 1150 1155
Asn Lys Phe Glu Ile Leu Glu Asn Glu Phe Leu Glu Asn Val Lys
1160 1165 1170
Ala Met Leu Glu Tyr Ser Glu
1175 1180
<210> 586
<211> 1197
<212> PRT
<213> Leptotrichia wadei
<400> 586
Met Lys Val Thr Lys Ile Asp Gly Leu Ser His Lys Lys Phe Glu Asp
1 5 10 15
Glu Gly Lys Leu Val Lys Phe Arg Asn Asn Lys Asn Ile Asn Glu Ile
20 25 30
Lys Glu Arg Leu Lys Lys Leu Lys Glu Leu Lys Leu Asp Asn Tyr Ile
35 40 45
Lys Asn Pro Glu Asn Val Lys Asn Lys Asp Lys Asp Ala Glu Lys Glu
50 55 60
Thr Lys Ile Arg Arg Thr Asn Leu Lys Lys Tyr Phe Ser Glu Ile Ile
65 70 75 80
Leu Arg Lys Glu Asp Glu Lys Tyr Ile Leu Lys Lys Thr Lys Lys Phe
85 90 95
Lys Asp Ile Asn Gln Glu Ile Asp Tyr Tyr Asp Val Lys Ser Lys Lys
100 105 110
Asn Gln Gln Glu Ile Phe Asp Val Leu Lys Glu Ile Leu Glu Leu Lys
115 120 125
Ile Lys Glu Thr Glu Lys Glu Glu Ile Ile Thr Phe Asp Ser Glu Lys
130 135 140
Leu Lys Lys Val Phe Gly Glu Asp Phe Val Lys Lys Glu Ala Lys Ile
145 150 155 160
Lys Ala Ile Glu Lys Ser Leu Lys Ile Asn Lys Ala Asn Tyr Lys Lys
165 170 175
Asp Ser Ile Lys Ile Gly Asp Asp Lys Tyr Ser Asn Val Lys Gly Glu
180 185 190
Asn Lys Arg Ser Arg Ile Tyr Glu Tyr Tyr Lys Lys Ser Glu Asn Leu
195 200 205
Lys Lys Phe Glu Glu Asn Ile Arg Glu Ala Phe Glu Lys Leu Tyr Thr
210 215 220
Glu Glu Asn Ile Lys Glu Leu Tyr Ser Lys Ile Glu Glu Ile Leu Lys
225 230 235 240
Lys Thr His Leu Lys Ser Ile Val Arg Glu Phe Tyr Gln Asn Glu Ile
245 250 255
Ile Gly Glu Ser Glu Phe Ser Lys Lys Asn Gly Asp Gly Ile Ser Ile
260 265 270
Leu Tyr Asn Gln Ile Lys Asp Ser Ile Lys Lys Glu Glu Asn Phe Ile
275 280 285
Glu Phe Ile Glu Asn Thr Gly Asn Leu Glu Leu Lys Glu Leu Thr Lys
290 295 300
Ser Gln Ile Phe Tyr Lys Tyr Phe Leu Glu Asn Glu Glu Leu Asn Asp
305 310 315 320
Glu Asn Ile Lys Phe Ala Phe Cys Tyr Phe Val Glu Ile Glu Val Asn
325 330 335
Asn Leu Leu Lys Glu Asn Val Tyr Lys Ile Lys Arg Phe Asn Glu Ser
340 345 350
Asn Lys Lys Arg Ile Glu Asn Ile Phe Glu Tyr Gly Lys Leu Lys Lys
355 360 365
Leu Ile Val Tyr Lys Leu Glu Asn Lys Leu Asn Asn Tyr Val Arg Asn
370 375 380
Cys Gly Lys Tyr Asn Tyr His Met Glu Asn Gly Asp Ile Ala Thr Ser
385 390 395 400
Asp Ile Asn Met Arg Asn Arg Gln Thr Glu Ala Phe Leu Arg Ser Ile
405 410 415
Ile Gly Val Ser Ser Phe Gly Tyr Phe Ser Leu Arg Asn Ile Leu Gly
420 425 430
Val Asn Asp Asp Asp Phe Tyr Glu Thr Glu Glu Asp Leu Thr Lys Lys
435 440 445
Glu Arg Arg Asn Leu Glu Lys Ala Lys Glu Asp Ile Thr Ile Lys Asn
450 455 460
Thr Phe Asp Glu Val Val Val Lys Ser Phe Gln Lys Lys Gly Ile Tyr
465 470 475 480
Asn Ile Lys Glu Asn Leu Lys Met Phe Tyr Gly Asp Ser Phe Asp Asn
485 490 495
Ala Asp Lys Asp Glu Leu Lys Gln Phe Phe Val Asn Met Leu Asn Ala
500 505 510
Ile Thr Ser Ile Arg His Arg Val Val His Tyr Asn Met Asn Thr Asn
515 520 525
Ser Glu Asn Ile Phe Asn Phe Ser Gly Ile Glu Val Ser Lys Leu Leu
530 535 540
Lys Ser Ile Phe Glu Lys Glu Thr Asp Lys Arg Glu Leu Lys Leu Lys
545 550 555 560
Ile Phe Arg Gln Leu Asn Ser Ala Gly Val Phe Asp Tyr Trp Glu Asn
565 570 575
Arg Lys Ile Asp Lys Tyr Leu Glu Asn Ile Glu Phe Lys Phe Val Asn
580 585 590
Lys Asn Ile Pro Phe Val Pro Ser Phe Thr Lys Leu Tyr Asn Arg Ile
595 600 605
Asp Asn Leu Lys Gly Asn Asn Ala Leu Asn Leu Gly Tyr Ile Asn Ile
610 615 620
Pro Lys Arg Lys Glu Ala Arg Asp Ser Gln Ile Tyr Leu Leu Lys Asn
625 630 635 640
Ile Tyr Tyr Gly Glu Phe Val Glu Lys Phe Val Asn Asn Asn Asp Asn
645 650 655
Phe Glu Lys Ile Phe Arg Glu Ile Ile Glu Ile Asn Lys Lys Asp Gly
660 665 670
Thr Asn Thr Lys Thr Lys Phe Tyr Lys Leu Glu Lys Phe Glu Thr Leu
675 680 685
Lys Ala Asn Ala Pro Ile Glu Tyr Leu Glu Lys Leu Gln Ser Leu His
690 695 700
Gln Ile Asn Tyr Asn Arg Glu Lys Val Glu Glu Asp Lys Asp Ile Tyr
705 710 715 720
Val Asp Phe Val Gln Lys Ile Phe Leu Lys Gly Phe Ile Asn Tyr Leu
725 730 735
Gln Gly Ser Asp Leu Leu Lys Ser Leu Asn Leu Leu Asn Leu Lys Lys
740 745 750
Asp Glu Ala Ile Ala Asn Lys Lys Ser Phe Tyr Asp Glu Lys Leu Lys
755 760 765
Leu Trp Gln Asn Asn Gly Ser Asn Leu Ser Lys Met Pro Glu Glu Ile
770 775 780
Tyr Asp Tyr Ile Lys Lys Ile Lys Ile Asn Lys Ile Asn Tyr Ser Asp
785 790 795 800
Arg Met Ser Ile Phe Tyr Leu Leu Leu Lys Leu Ile Asp His Lys Glu
805 810 815
Leu Thr Asn Leu Arg Gly Asn Leu Glu Lys Tyr Val Ser Met Asn Lys
820 825 830
Asn Lys Ile Tyr Ser Glu Glu Leu Asn Ile Val Asn Leu Val Ser Leu
835 840 845
Asp Asn Asn Lys Val Arg Ala Asn Phe Asn Leu Lys Pro Glu Asp Ile
850 855 860
Gly Lys Phe Leu Lys Thr Glu Thr Ser Ile Arg Asn Ile Asn Gln Leu
865 870 875 880
Asn Asn Phe Ser Glu Ile Phe Ala Asp Gly Glu Asn Val Ile Lys His
885 890 895
Arg Ser Phe Tyr Asn Ile Lys Lys Tyr Gly Ile Leu Asp Leu Leu Glu
900 905 910
Lys Ile Val Asp Lys Ala Asp Leu Lys Ile Thr Lys Glu Glu Ile Lys
915 920 925
Lys Tyr Glu Asn Leu Gln Asn Glu Leu Lys Arg Asn Asp Phe Tyr Lys
930 935 940
Ile Gln Glu Arg Ile His Arg Asn Tyr Asn Gln Lys Pro Phe Leu Ile
945 950 955 960
Lys Asn Asn Glu Lys Asp Phe Asn Asp Tyr Lys Lys Ala Ile Glu Asn
965 970 975
Ile Gln Asn Tyr Thr Gln Leu Lys Asn Lys Ile Glu Phe Asn Asp Leu
980 985 990
Asn Leu Leu Gln Ser Leu Leu Phe Arg Ile Leu His Arg Leu Ala Gly
995 1000 1005
Tyr Thr Ser Leu Trp Glu Arg Asp Leu Gln Phe Lys Leu Lys Gly
1010 1015 1020
Glu Tyr Pro Glu Asn Lys Tyr Ile Asp Glu Ile Phe Asn Phe Asp
1025 1030 1035
Asn Ser Lys Asn Lys Ile Tyr Asn Glu Lys Asn Glu Arg Gly Gly
1040 1045 1050
Ser Val Val Ser Lys Tyr Gly Tyr Phe Leu Val Glu Lys Asp Gly
1055 1060 1065
Glu Ile Gln Arg Lys Asn Ala Arg Asp Lys Lys Lys Asn Lys Ile
1070 1075 1080
Ile Lys Lys Glu Gly Leu Glu Ile Arg Asn Tyr Ile Ala His Phe
1085 1090 1095
Asn Tyr Ile Pro Asp Ala Thr Lys Ser Ile Leu Glu Ile Leu Glu
1100 1105 1110
Glu Leu Arg Asn Leu Leu Lys Tyr Asp Arg Lys Leu Lys Asn Ala
1115 1120 1125
Val Met Lys Ser Ile Lys Asp Ile Phe Lys Glu Tyr Gly Leu Ile
1130 1135 1140
Ile Glu Phe Lys Ile Ser His Val Asn Asn Ser Glu Lys Ile Glu
1145 1150 1155
Val Leu Asn Val Asp Ser Glu Lys Ile Lys His Leu Lys Asn Asn
1160 1165 1170
Gly Leu Val Thr Thr Arg Asn Ser Glu Asp Leu Cys Glu Leu Ile
1175 1180 1185
Lys Met Met Leu Glu Tyr Lys Lys Ser
1190 1195
<210> 587
<211> 1152
<212> PRT
<213> Leptotrichia wadei
<400> 587
Met Lys Val Thr Lys Val Asp Gly Ile Ser His Lys Lys Tyr Ile Glu
1 5 10 15
Glu Gly Lys Leu Val Lys Ser Thr Ser Glu Glu Asn Arg Thr Ser Glu
20 25 30
Arg Leu Ser Glu Leu Leu Ser Ile Arg Leu Asp Ile Tyr Ile Lys Asn
35 40 45
Pro Asp Asn Ala Ser Glu Glu Glu Asn Arg Ile Arg Arg Glu Asn Leu
50 55 60
Lys Lys Phe Phe Ser Asn Lys Val Leu His Leu Lys Asp Ser Val Leu
65 70 75 80
Tyr Leu Lys Asn Arg Lys Glu Lys Asn Ala Val Gln Asp Lys Asn Tyr
85 90 95
Ser Glu Glu Asp Ile Ser Glu Tyr Asp Leu Lys Asn Lys Asn Ser Phe
100 105 110
Ser Val Leu Lys Lys Ile Leu Leu Asn Glu Asp Val Asn Ser Glu Glu
115 120 125
Leu Glu Ile Phe Arg Lys Asp Val Glu Ala Lys Leu Asn Lys Ile Asn
130 135 140
Ser Leu Lys Tyr Ser Phe Glu Glu Asn Lys Ala Asn Tyr Gln Lys Ile
145 150 155 160
Asn Glu Asn Asn Val Glu Lys Val Gly Gly Lys Ser Lys Arg Asn Ile
165 170 175
Ile Tyr Asp Tyr Tyr Arg Glu Ser Ala Lys Arg Asn Asp Tyr Ile Asn
180 185 190
Asn Val Gln Glu Ala Phe Asp Lys Leu Tyr Lys Lys Glu Asp Ile Glu
195 200 205
Lys Leu Phe Phe Leu Ile Glu Asn Ser Lys Lys His Glu Lys Tyr Lys
210 215 220
Ile Arg Glu Tyr Tyr His Lys Ile Ile Gly Arg Lys Asn Asp Lys Glu
225 230 235 240
Asn Phe Ala Lys Ile Ile Tyr Glu Glu Ile Gln Asn Val Asn Asn Ile
245 250 255
Lys Glu Leu Ile Glu Lys Ile Pro Asp Met Ser Glu Leu Lys Lys Ser
260 265 270
Gln Val Phe Tyr Lys Tyr Tyr Leu Asp Lys Glu Glu Leu Asn Asp Lys
275 280 285
Asn Ile Lys Tyr Ala Phe Cys His Phe Val Glu Ile Glu Met Ser Gln
290 295 300
Leu Leu Lys Asn Tyr Val Tyr Lys Arg Leu Ser Asn Ile Ser Asn Asp
305 310 315 320
Lys Ile Lys Arg Ile Phe Glu Tyr Gln Asn Leu Lys Lys Leu Ile Glu
325 330 335
Asn Lys Leu Leu Asn Lys Leu Asp Thr Tyr Val Arg Asn Cys Gly Lys
340 345 350
Tyr Asn Tyr Tyr Leu Gln Val Gly Glu Ile Ala Thr Ser Asp Phe Ile
355 360 365
Ala Arg Asn Arg Gln Asn Glu Ala Phe Leu Arg Asn Ile Ile Gly Val
370 375 380
Ser Ser Val Ala Tyr Phe Ser Leu Arg Asn Ile Leu Glu Thr Glu Asn
385 390 395 400
Glu Asn Asp Ile Thr Gly Arg Met Arg Gly Lys Thr Val Lys Asn Asn
405 410 415
Lys Gly Glu Glu Lys Tyr Val Ser Gly Glu Val Asp Lys Ile Tyr Asn
420 425 430
Glu Asn Lys Gln Asn Glu Val Lys Glu Asn Leu Lys Met Phe Tyr Ser
435 440 445
Tyr Asp Phe Asn Met Asp Asn Lys Asn Glu Ile Glu Asp Phe Phe Ala
450 455 460
Asn Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe
465 470 475 480
Asn Leu Glu Leu Glu Gly Lys Asp Ile Phe Ala Phe Lys Asn Ile Ala
485 490 495
Pro Ser Glu Ile Ser Lys Lys Met Phe Gln Asn Glu Ile Asn Glu Lys
500 505 510
Lys Leu Lys Leu Lys Ile Phe Lys Gln Leu Asn Ser Ala Asn Val Phe
515 520 525
Asn Tyr Tyr Glu Lys Asp Val Ile Ile Lys Tyr Leu Lys Asn Thr Lys
530 535 540
Phe Asn Phe Val Asn Lys Asn Ile Pro Phe Val Pro Ser Phe Thr Lys
545 550 555 560
Leu Tyr Asn Lys Ile Glu Asp Leu Arg Asn Thr Leu Lys Phe Phe Trp
565 570 575
Ser Val Pro Lys Asp Lys Glu Glu Lys Asp Ala Gln Ile Tyr Leu Leu
580 585 590
Lys Asn Ile Tyr Tyr Gly Glu Phe Leu Asn Lys Phe Val Lys Asn Ser
595 600 605
Lys Val Phe Phe Lys Ile Thr Asn Glu Val Ile Lys Ile Asn Lys Gln
610 615 620
Arg Asn Gln Lys Thr Gly His Tyr Lys Tyr Gln Lys Phe Glu Asn Ile
625 630 635 640
Glu Lys Thr Val Pro Val Glu Tyr Leu Ala Ile Ile Gln Ser Arg Glu
645 650 655
Met Ile Asn Asn Gln Asp Lys Glu Glu Lys Asn Thr Tyr Ile Asp Phe
660 665 670
Ile Gln Gln Ile Phe Leu Lys Gly Phe Ile Asp Tyr Leu Asn Lys Asn
675 680 685
Asn Leu Lys Tyr Ile Glu Ser Asn Asn Asn Asn Asp Asn Asn Asp Ile
690 695 700
Phe Ser Lys Ile Lys Ile Lys Lys Asp Asn Lys Glu Lys Tyr Asp Lys
705 710 715 720
Ile Leu Lys Asn Tyr Glu Lys His Asn Arg Asn Lys Glu Ile Pro His
725 730 735
Glu Ile Asn Glu Phe Val Arg Glu Ile Lys Leu Gly Lys Ile Leu Lys
740 745 750
Tyr Thr Glu Asn Leu Asn Met Phe Tyr Leu Ile Leu Lys Leu Leu Asn
755 760 765
His Lys Glu Leu Thr Asn Leu Lys Gly Ser Leu Glu Lys Tyr Gln Ser
770 775 780
Ala Asn Lys Glu Glu Thr Phe Ser Asp Glu Leu Glu Leu Ile Asn Leu
785 790 795 800
Leu Asn Leu Asp Asn Asn Arg Val Thr Glu Asp Phe Glu Leu Glu Ala
805 810 815
Asn Glu Ile Gly Lys Phe Leu Asp Phe Asn Glu Asn Lys Ile Lys Asp
820 825 830
Arg Lys Glu Leu Lys Lys Phe Asp Thr Asn Lys Ile Tyr Phe Asp Gly
835 840 845
Glu Asn Ile Ile Lys His Arg Ala Phe Tyr Asn Ile Lys Lys Tyr Gly
850 855 860
Met Leu Asn Leu Leu Glu Lys Ile Ala Asp Lys Ala Lys Tyr Lys Ile
865 870 875 880
Ser Leu Lys Glu Leu Lys Glu Tyr Ser Asn Lys Lys Asn Glu Ile Glu
885 890 895
Lys Asn Tyr Thr Met Gln Gln Asn Leu His Arg Lys Tyr Ala Arg Pro
900 905 910
Lys Lys Asp Glu Lys Phe Asn Asp Glu Asp Tyr Lys Glu Tyr Glu Lys
915 920 925
Ala Ile Gly Asn Ile Gln Lys Tyr Thr His Leu Lys Asn Lys Val Glu
930 935 940
Phe Asn Glu Leu Asn Leu Leu Gln Gly Leu Leu Leu Lys Ile Leu His
945 950 955 960
Arg Leu Val Gly Tyr Thr Ser Ile Trp Glu Arg Asp Leu Arg Phe Arg
965 970 975
Leu Lys Gly Glu Phe Pro Glu Asn His Tyr Ile Glu Glu Ile Phe Asn
980 985 990
Phe Asp Asn Ser Lys Asn Val Lys Tyr Lys Ser Gly Gln Ile Val Glu
995 1000 1005
Lys Tyr Ile Asn Phe Tyr Lys Glu Leu Tyr Lys Asp Asn Val Glu
1010 1015 1020
Lys Arg Ser Ile Tyr Ser Asp Lys Lys Val Lys Lys Leu Lys Gln
1025 1030 1035
Glu Lys Lys Asp Leu Tyr Ile Arg Asn Tyr Ile Ala His Phe Asn
1040 1045 1050
Tyr Ile Pro His Ala Glu Ile Ser Leu Leu Glu Val Leu Glu Asn
1055 1060 1065
Leu Arg Lys Leu Leu Ser Tyr Asp Arg Lys Leu Lys Asn Ala Ile
1070 1075 1080
Met Lys Ser Ile Val Asp Ile Leu Lys Glu Tyr Gly Phe Val Ala
1085 1090 1095
Thr Phe Lys Ile Gly Ala Asp Lys Lys Ile Glu Ile Gln Thr Leu
1100 1105 1110
Glu Ser Glu Lys Ile Val His Leu Lys Asn Leu Lys Lys Lys Lys
1115 1120 1125
Leu Met Thr Asp Arg Asn Ser Glu Glu Leu Cys Glu Leu Val Lys
1130 1135 1140
Val Met Phe Glu Tyr Lys Ala Leu Glu
1145 1150
<210> 588
<211> 1159
<212> PRT
<213> Leptotrichia buccalis
<400> 588
Met Lys Val Thr Lys Val Gly Gly Ile Ser His Lys Lys Tyr Thr Ser
1 5 10 15
Glu Gly Arg Leu Val Lys Ser Glu Ser Glu Glu Asn Arg Thr Asp Glu
20 25 30
Arg Leu Ser Ala Leu Leu Asn Met Arg Leu Asp Met Tyr Ile Lys Asn
35 40 45
Pro Ser Ser Thr Glu Thr Lys Glu Asn Gln Lys Arg Ile Gly Lys Leu
50 55 60
Lys Lys Phe Phe Ser Asn Lys Met Val Tyr Leu Lys Asp Asn Thr Leu
65 70 75 80
Ser Leu Lys Asn Gly Lys Lys Glu Asn Ile Asp Arg Glu Tyr Ser Glu
85 90 95
Thr Asp Ile Leu Glu Ser Asp Val Arg Asp Lys Lys Asn Phe Ala Val
100 105 110
Leu Lys Lys Ile Tyr Leu Asn Glu Asn Val Asn Ser Glu Glu Leu Glu
115 120 125
Val Phe Arg Asn Asp Ile Lys Lys Lys Leu Asn Lys Ile Asn Ser Leu
130 135 140
Lys Tyr Ser Phe Glu Lys Asn Lys Ala Asn Tyr Gln Lys Ile Asn Glu
145 150 155 160
Asn Asn Ile Glu Lys Val Glu Gly Lys Ser Lys Arg Asn Ile Ile Tyr
165 170 175
Asp Tyr Tyr Arg Glu Ser Ala Lys Arg Asp Ala Tyr Val Ser Asn Val
180 185 190
Lys Glu Ala Phe Asp Lys Leu Tyr Lys Glu Glu Asp Ile Ala Lys Leu
195 200 205
Val Leu Glu Ile Glu Asn Leu Thr Lys Leu Glu Lys Tyr Lys Ile Arg
210 215 220
Glu Phe Tyr His Glu Ile Ile Gly Arg Lys Asn Asp Lys Glu Asn Phe
225 230 235 240
Ala Lys Ile Ile Tyr Glu Glu Ile Gln Asn Val Asn Asn Met Lys Glu
245 250 255
Leu Ile Glu Lys Val Pro Asp Met Ser Glu Leu Lys Lys Ser Gln Val
260 265 270
Phe Tyr Lys Tyr Tyr Leu Asp Lys Glu Glu Leu Asn Asp Lys Asn Ile
275 280 285
Lys Tyr Ala Phe Cys His Phe Val Glu Ile Glu Met Ser Gln Leu Leu
290 295 300
Lys Asn Tyr Val Tyr Lys Arg Leu Ser Asn Ile Ser Asn Asp Lys Ile
305 310 315 320
Lys Arg Ile Phe Glu Tyr Gln Asn Leu Lys Lys Leu Ile Glu Asn Lys
325 330 335
Leu Leu Asn Lys Leu Asp Thr Tyr Val Arg Asn Cys Gly Lys Tyr Asn
340 345 350
Tyr Tyr Leu Gln Asp Gly Glu Ile Ala Thr Ser Asp Phe Ile Ala Arg
355 360 365
Asn Arg Gln Asn Glu Ala Phe Leu Arg Asn Ile Ile Gly Val Ser Ser
370 375 380
Val Ala Tyr Phe Ser Leu Arg Asn Ile Leu Glu Thr Glu Asn Glu Asn
385 390 395 400
Asp Ile Thr Gly Arg Met Arg Gly Lys Thr Val Lys Asn Asn Lys Gly
405 410 415
Glu Glu Lys Tyr Val Ser Gly Glu Val Asp Lys Ile Tyr Asn Glu Asn
420 425 430
Lys Lys Asn Glu Val Lys Glu Asn Leu Lys Met Phe Tyr Ser Tyr Asp
435 440 445
Phe Asn Met Asp Asn Lys Asn Glu Ile Glu Asp Phe Phe Ala Asn Ile
450 455 460
Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe Asn Leu
465 470 475 480
Glu Leu Glu Gly Lys Asp Ile Phe Ala Phe Lys Asn Ile Ala Pro Ser
485 490 495
Glu Ile Ser Lys Lys Met Phe Gln Asn Glu Ile Asn Glu Lys Lys Leu
500 505 510
Lys Leu Lys Ile Phe Arg Gln Leu Asn Ser Ala Asn Val Phe Arg Tyr
515 520 525
Leu Glu Lys Tyr Lys Ile Leu Asn Tyr Leu Lys Arg Thr Arg Phe Glu
530 535 540
Phe Val Asn Lys Asn Ile Pro Phe Val Pro Ser Phe Thr Lys Leu Tyr
545 550 555 560
Ser Arg Ile Asp Asp Leu Lys Asn Ser Leu Gly Ile Tyr Trp Lys Thr
565 570 575
Pro Lys Thr Asn Asp Asp Asn Lys Thr Lys Glu Ile Ile Asp Ala Gln
580 585 590
Ile Tyr Leu Leu Lys Asn Ile Tyr Tyr Gly Glu Phe Leu Asn Tyr Phe
595 600 605
Met Ser Asn Asn Gly Asn Phe Phe Glu Ile Ser Lys Glu Ile Ile Glu
610 615 620
Leu Asn Lys Asn Asp Lys Arg Asn Leu Lys Thr Gly Phe Tyr Lys Leu
625 630 635 640
Gln Lys Phe Glu Asp Ile Gln Glu Lys Ile Pro Lys Glu Tyr Leu Ala
645 650 655
Asn Ile Gln Ser Leu Tyr Met Ile Asn Ala Gly Asn Gln Asp Glu Glu
660 665 670
Glu Lys Asp Thr Tyr Ile Asp Phe Ile Gln Lys Ile Phe Leu Lys Gly
675 680 685
Phe Met Thr Tyr Leu Ala Asn Asn Gly Arg Leu Ser Leu Ile Tyr Ile
690 695 700
Gly Ser Asp Glu Glu Thr Asn Thr Ser Leu Ala Glu Lys Lys Gln Glu
705 710 715 720
Phe Asp Lys Phe Leu Lys Lys Tyr Glu Gln Asn Asn Asn Ile Lys Ile
725 730 735
Pro Tyr Glu Ile Asn Glu Phe Leu Arg Glu Ile Lys Leu Gly Asn Ile
740 745 750
Leu Lys Tyr Thr Glu Arg Leu Asn Met Phe Tyr Leu Ile Leu Lys Leu
755 760 765
Leu Asn His Lys Glu Leu Thr Asn Leu Lys Gly Ser Leu Glu Lys Tyr
770 775 780
Gln Ser Ala Asn Lys Glu Glu Ala Phe Ser Asp Gln Leu Glu Leu Ile
785 790 795 800
Asn Leu Leu Asn Leu Asp Asn Asn Arg Val Thr Glu Asp Phe Glu Leu
805 810 815
Glu Ala Asp Glu Ile Gly Lys Phe Leu Asp Phe Asn Gly Asn Lys Val
820 825 830
Lys Asp Asn Lys Glu Leu Lys Lys Phe Asp Thr Asn Lys Ile Tyr Phe
835 840 845
Asp Gly Glu Asn Ile Ile Lys His Arg Ala Phe Tyr Asn Ile Lys Lys
850 855 860
Tyr Gly Met Leu Asn Leu Leu Glu Lys Ile Ala Asp Lys Ala Gly Tyr
865 870 875 880
Lys Ile Ser Ile Glu Glu Leu Lys Lys Tyr Ser Asn Lys Lys Asn Glu
885 890 895
Ile Glu Lys Asn His Lys Met Gln Glu Asn Leu His Arg Lys Tyr Ala
900 905 910
Arg Pro Arg Lys Asp Glu Lys Phe Thr Asp Glu Asp Tyr Glu Ser Tyr
915 920 925
Lys Gln Ala Ile Glu Asn Ile Glu Glu Tyr Thr His Leu Lys Asn Lys
930 935 940
Val Glu Phe Asn Glu Leu Asn Leu Leu Gln Gly Leu Leu Leu Arg Ile
945 950 955 960
Leu His Arg Leu Val Gly Tyr Thr Ser Ile Trp Glu Arg Asp Leu Arg
965 970 975
Phe Arg Leu Lys Gly Glu Phe Pro Glu Asn Gln Tyr Ile Glu Glu Ile
980 985 990
Phe Asn Phe Glu Asn Lys Lys Asn Val Lys Tyr Lys Gly Gly Gln Ile
995 1000 1005
Val Glu Lys Tyr Ile Lys Phe Tyr Lys Glu Leu His Gln Asn Asp
1010 1015 1020
Glu Val Lys Ile Asn Lys Tyr Ser Ser Ala Asn Ile Lys Val Leu
1025 1030 1035
Lys Gln Glu Lys Lys Asp Leu Tyr Ile Arg Asn Tyr Ile Ala His
1040 1045 1050
Phe Asn Tyr Ile Pro His Ala Glu Ile Ser Leu Leu Glu Val Leu
1055 1060 1065
Glu Asn Leu Arg Lys Leu Leu Ser Tyr Asp Arg Lys Leu Lys Asn
1070 1075 1080
Ala Val Met Lys Ser Val Val Asp Ile Leu Lys Glu Tyr Gly Phe
1085 1090 1095
Val Ala Thr Phe Lys Ile Gly Ala Asp Lys Lys Ile Gly Ile Gln
1100 1105 1110
Thr Leu Glu Ser Glu Lys Ile Val His Leu Lys Asn Leu Lys Lys
1115 1120 1125
Lys Lys Leu Met Thr Asp Arg Asn Ser Glu Glu Leu Cys Lys Leu
1130 1135 1140
Val Lys Ile Met Phe Glu Tyr Lys Met Glu Glu Lys Lys Ser Glu
1145 1150 1155
Asn
<210> 589
<211> 913
<212> PRT
<213> Leptotrichia sp.
<400> 589
Met Lys Glu Leu Ile Glu Lys Val Pro Asn Val Ser Glu Leu Lys Lys
1 5 10 15
Ser Gln Val Phe Tyr Lys Tyr Tyr Leu Asn Lys Glu Lys Leu Asn Asp
20 25 30
Glu Asn Ile Lys Tyr Val Phe Cys His Phe Val Glu Ile Glu Met Ser
35 40 45
Lys Leu Leu Lys Asn Tyr Val Tyr Lys Lys Pro Ser Asn Ile Ser Asn
50 55 60
Asp Lys Val Lys Arg Ile Phe Glu Tyr Gln Ser Leu Lys Lys Leu Ile
65 70 75 80
Glu Asn Lys Leu Leu Asn Lys Leu Asp Thr Tyr Ile Arg Asn Cys Gly
85 90 95
Lys Tyr Ser Phe Tyr Leu Gln Asp Gly Glu Ile Ala Thr Ser Asp Phe
100 105 110
Ile Val Gly Asn Arg Gln Asn Glu Ala Phe Leu Arg Asn Ile Ile Gly
115 120 125
Val Ser Ser Ala Ala Tyr Phe Ser Leu Arg Asn Ile Leu Glu Thr Glu
130 135 140
Asn Glu Asn Asp Ile Thr Gly Lys Met Arg Gly Lys Thr Val Lys Asn
145 150 155 160
Lys Lys Gly Glu Glu Lys Tyr Ile Ser Gly Glu Ile Asp Lys Leu Tyr
165 170 175
Asp Asn Asn Lys Gln Asn Glu Val Lys Lys Asn Leu Lys Met Phe Tyr
180 185 190
Ser Tyr Asp Phe Asn Met Asn Ser Lys Lys Glu Ile Glu Asp Phe Phe
195 200 205
Ser Asn Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His
210 215 220
Phe Asn Leu Glu Leu Glu Gly Lys Asp Ile Phe Thr Phe Lys Asn Ile
225 230 235 240
Val Pro Ser Gln Ile Ser Lys Lys Met Phe His Asp Glu Ile Asn Glu
245 250 255
Lys Lys Leu Lys Leu Lys Ile Phe Lys Gln Leu Asn Ser Ala Asn Val
260 265 270
Phe Arg Tyr Leu Glu Lys Tyr Lys Ile Leu Asn Tyr Leu Asn Arg Thr
275 280 285
Arg Phe Glu Phe Val Asn Lys Asn Ile Pro Phe Val Pro Ser Phe Thr
290 295 300
Lys Leu Tyr Ser Arg Ile Asp Asp Leu Lys Asn Ser Leu Cys Ile Tyr
305 310 315 320
Trp Lys Ile Pro Lys Ala Asn Asp Asn Asn Lys Thr Lys Glu Ile Thr
325 330 335
Asp Ala Gln Ile Tyr Leu Leu Lys Asn Ile Tyr Tyr Ser Glu Phe Leu
340 345 350
Asn Tyr Phe Met Ser Asn Asn Gly Asn Phe Phe Glu Ile Ile Lys Glu
355 360 365
Ile Ile Glu Leu Asn Lys Asn Asp Lys Arg Asn Leu Lys Thr Gly Phe
370 375 380
Tyr Lys Leu Gln Lys Phe Glu Asn Leu Gln Glu Lys Thr Pro Lys Glu
385 390 395 400
Tyr Leu Ala Asn Ile Gln Ser Phe Tyr Met Ile Asp Ala Gly Asn Lys
405 410 415
Asp Glu Glu Glu Lys Asp Ala Tyr Ile Asp Phe Ile Gln Lys Ile Phe
420 425 430
Leu Lys Gly Phe Met Thr Tyr Leu Ala Asn Asn Gly Arg Leu Ser Leu
435 440 445
Met Tyr Ile Gly Asn Asp Glu Gln Ile Asn Thr Ser Leu Ala Glu Lys
450 455 460
Lys Gln Glu Phe Asp Lys Phe Leu Lys Lys Tyr Glu Gln Asn Asn Asn
465 470 475 480
Ile Lys Ile Pro Tyr Glu Ile Asn Glu Phe Leu Arg Glu Ile Lys Leu
485 490 495
Gly Asn Ile Leu Lys Tyr Thr Glu Arg Leu Asn Met Phe Tyr Leu Ile
500 505 510
Leu Lys Leu Leu Asn His Lys Glu Leu Thr Asn Leu Lys Gly Ser Leu
515 520 525
Glu Lys Tyr Gln Ser Ala Asn Lys Glu Glu Ala Phe Ser Asp Gln Leu
530 535 540
Glu Leu Ile Asn Leu Leu Asn Leu Asp Asn Asn Arg Val Thr Glu Asp
545 550 555 560
Phe Glu Leu Glu Ala Asp Glu Ile Gly Lys Phe Leu Asp Phe Asn Gly
565 570 575
Asn Lys Val Lys Asp Asn Lys Glu Leu Lys Lys Phe Asp Thr Asn Lys
580 585 590
Ile Tyr Phe Asp Gly Glu Asn Ile Ile Lys His Arg Ala Phe Tyr Asn
595 600 605
Ile Lys Lys Tyr Gly Met Leu Asn Leu Leu Glu Lys Ile Ser Asp Glu
610 615 620
Ala Lys Tyr Lys Ile Ser Ile Glu Glu Leu Lys Asn Tyr Ser Asn Lys
625 630 635 640
Lys Asn Glu Ile Glu Lys Asn His Thr Asn Gln Glu Asn Leu His Arg
645 650 655
Lys Tyr Ala Arg Pro Arg Lys Asp Glu Lys Phe Asn Asp Glu Asp Tyr
660 665 670
Lys Lys Tyr Glu Lys Ala Ile Arg Asn Ile Gln Gln Tyr Thr His Leu
675 680 685
Lys Asn Lys Val Glu Phe Asn Glu Leu Asn Leu Leu Gln Ser Leu Leu
690 695 700
Leu Arg Ile Leu His Arg Leu Val Gly Tyr Thr Ser Ile Trp Glu Arg
705 710 715 720
Asp Leu Arg Phe Arg Leu Lys Gly Glu Phe Pro Glu Asn Gln Tyr Ile
725 730 735
Glu Glu Ile Phe Asn Phe Asn Asn Ser Lys Asn Val Lys Tyr Lys Asn
740 745 750
Gly Gln Ile Val Glu Lys Tyr Ile Ser Phe Tyr Lys Glu Leu Tyr Lys
755 760 765
Asp Asp Thr Glu Lys Ile Ser Ile Tyr Ser Asp Lys Lys Val Lys Glu
770 775 780
Leu Lys Lys Glu Lys Lys Asp Leu Tyr Ile Arg Asn Tyr Ile Ala His
785 790 795 800
Phe Asn Tyr Ile Pro Asn Ala Glu Ile Ser Leu Leu Glu Val Leu Glu
805 810 815
Asn Leu Arg Lys Leu Leu Ser Tyr Asp Arg Lys Leu Lys Asn Ala Ile
820 825 830
Met Lys Ser Ile Val Asp Ile Leu Lys Glu Tyr Gly Phe Val Val Thr
835 840 845
Phe Lys Ile Glu Lys Asp Lys Lys Ile Arg Ile Glu Ser Leu Lys Ser
850 855 860
Glu Glu Val Val His Leu Lys Lys Leu Lys Leu Lys Asp Asn Asp Lys
865 870 875 880
Lys Lys Glu Pro Ile Lys Thr Tyr Arg Asn Ser Lys Glu Leu Cys Glu
885 890 895
Leu Val Lys Val Met Phe Glu Tyr Lys Met Lys Glu Lys Lys Ser Glu
900 905 910
Asn
<210> 590
<211> 1385
<212> PRT
<213> Leptotrichia sp.
<400> 590
Met Gly Asn Leu Phe Gly His Lys Arg Trp Tyr Glu Val Arg Asp Lys
1 5 10 15
Lys Asp Phe Lys Ile Lys Arg Lys Val Lys Val Lys Arg Asn Tyr Asp
20 25 30
Gly Asn Lys Tyr Ile Leu Asn Ile Asn Glu Asn Asn Asn Lys Glu Lys
35 40 45
Ile Asp Asn Asn Lys Phe Ile Gly Glu Phe Val Asn Tyr Lys Lys Asn
50 55 60
Asn Asn Val Leu Lys Glu Phe Lys Arg Lys Phe His Ala Gly Asn Ile
65 70 75 80
Leu Phe Lys Leu Lys Gly Lys Glu Glu Ile Ile Arg Ile Glu Asn Asn
85 90 95
Asp Asp Phe Leu Glu Thr Glu Glu Val Val Leu Tyr Ile Glu Val Tyr
100 105 110
Gly Lys Ser Glu Lys Leu Lys Ala Leu Glu Ile Thr Lys Lys Lys Ile
115 120 125
Ile Asp Glu Ala Ile Arg Gln Gly Ile Thr Lys Asp Asp Lys Lys Ile
130 135 140
Glu Ile Lys Arg Gln Glu Asn Glu Glu Glu Ile Glu Ile Asp Ile Arg
145 150 155 160
Asp Glu Tyr Thr Asn Lys Thr Leu Asn Asp Cys Ser Ile Ile Leu Arg
165 170 175
Ile Ile Glu Asn Asp Glu Leu Glu Thr Lys Lys Ser Ile Tyr Glu Ile
180 185 190
Phe Lys Asn Ile Asn Met Ser Leu Tyr Lys Ile Ile Glu Lys Ile Ile
195 200 205
Glu Asn Glu Thr Glu Lys Val Phe Glu Asn Arg Tyr Tyr Glu Glu His
210 215 220
Leu Arg Glu Lys Leu Leu Lys Asp Asn Lys Ile Asp Val Ile Leu Thr
225 230 235 240
Asn Phe Met Glu Ile Arg Glu Lys Ile Lys Ser Asn Leu Glu Ile Met
245 250 255
Gly Phe Val Lys Phe Tyr Leu Asn Val Ser Gly Asp Lys Lys Lys Ser
260 265 270
Glu Asn Lys Lys Met Phe Val Glu Lys Ile Leu Asn Thr Asn Val Asp
275 280 285
Leu Thr Val Glu Asp Ile Val Asp Phe Ile Val Lys Glu Leu Lys Phe
290 295 300
Trp Asn Ile Thr Lys Arg Ile Glu Lys Val Lys Lys Phe Asn Asn Glu
305 310 315 320
Phe Leu Glu Asn Arg Arg Asn Arg Thr Tyr Ile Lys Ser Tyr Val Leu
325 330 335
Leu Asp Lys His Glu Lys Phe Lys Ile Glu Arg Glu Asn Lys Lys Asp
340 345 350
Lys Ile Val Lys Phe Phe Val Glu Asn Ile Lys Asn Asn Ser Ile Lys
355 360 365
Glu Lys Ile Glu Lys Ile Leu Ala Glu Phe Lys Ile Asn Glu Leu Ile
370 375 380
Lys Lys Leu Glu Lys Glu Leu Lys Lys Gly Asn Cys Asp Thr Glu Ile
385 390 395 400
Phe Gly Ile Phe Lys Lys His Tyr Lys Val Asn Phe Asp Ser Lys Lys
405 410 415
Phe Ser Asn Lys Ser Asp Glu Glu Lys Glu Leu Tyr Lys Ile Ile Tyr
420 425 430
Arg Tyr Leu Lys Gly Arg Ile Glu Lys Ile Leu Val Asn Glu Gln Lys
435 440 445
Val Arg Leu Lys Lys Met Glu Lys Ile Glu Ile Glu Lys Ile Leu Asn
450 455 460
Glu Ser Ile Leu Ser Glu Lys Ile Leu Lys Arg Val Lys Gln Tyr Thr
465 470 475 480
Leu Glu His Ile Met Tyr Leu Gly Lys Leu Arg His Asn Asp Ile Val
485 490 495
Lys Met Thr Val Asn Thr Asp Asp Phe Ser Arg Leu His Ala Lys Glu
500 505 510
Glu Leu Asp Leu Glu Leu Ile Thr Phe Phe Ala Ser Thr Asn Met Glu
515 520 525
Leu Asn Lys Ile Phe Asn Gly Lys Glu Lys Val Thr Asp Phe Phe Gly
530 535 540
Phe Asn Leu Asn Gly Gln Lys Ile Thr Leu Lys Glu Lys Val Pro Ser
545 550 555 560
Phe Lys Leu Asn Ile Leu Lys Lys Leu Asn Phe Ile Asn Asn Glu Asn
565 570 575
Asn Ile Asp Glu Lys Leu Ser His Phe Tyr Ser Phe Gln Lys Glu Gly
580 585 590
Tyr Leu Leu Arg Asn Lys Ile Leu His Asn Ser Tyr Gly Asn Ile Gln
595 600 605
Glu Thr Lys Asn Leu Lys Gly Glu Tyr Glu Asn Val Glu Lys Leu Ile
610 615 620
Lys Glu Leu Lys Val Ser Asp Glu Glu Ile Ser Lys Ser Leu Ser Leu
625 630 635 640
Asp Val Ile Phe Glu Gly Lys Val Asp Ile Ile Asn Lys Ile Asn Ser
645 650 655
Leu Lys Ile Gly Glu Tyr Lys Asp Lys Lys Tyr Leu Pro Ser Phe Ser
660 665 670
Lys Ile Val Leu Glu Ile Thr Arg Lys Phe Arg Glu Ile Asn Lys Asp
675 680 685
Lys Leu Phe Asp Ile Glu Ser Glu Lys Ile Ile Leu Asn Ala Val Lys
690 695 700
Tyr Val Asn Lys Ile Leu Tyr Glu Lys Ile Thr Ser Asn Glu Glu Asn
705 710 715 720
Glu Phe Leu Lys Thr Leu Pro Asp Lys Leu Val Lys Lys Ser Asn Asn
725 730 735
Lys Lys Glu Asn Lys Asn Leu Leu Ser Ile Glu Glu Tyr Tyr Lys Asn
740 745 750
Ala Gln Val Ser Ser Ser Lys Gly Asp Lys Lys Ala Ile Lys Lys Tyr
755 760 765
Gln Asn Lys Val Thr Asn Ala Tyr Leu Glu Tyr Leu Glu Asn Thr Phe
770 775 780
Thr Glu Ile Ile Asp Phe Ser Lys Phe Asn Leu Asn Tyr Asp Glu Ile
785 790 795 800
Lys Thr Lys Ile Glu Glu Arg Lys Asp Asn Lys Ser Lys Ile Ile Ile
805 810 815
Asp Ser Ile Ser Thr Asn Ile Asn Ile Thr Asn Asp Ile Glu Tyr Ile
820 825 830
Ile Ser Ile Phe Ala Leu Leu Asn Ser Asn Thr Tyr Ile Asn Lys Ile
835 840 845
Arg Asn Arg Phe Phe Ala Thr Ser Val Trp Leu Glu Lys Gln Asn Gly
850 855 860
Thr Lys Glu Tyr Asp Tyr Glu Asn Ile Ile Ser Ile Leu Asp Glu Val
865 870 875 880
Leu Leu Ile Asn Leu Leu Arg Glu Asn Asn Ile Thr Asp Ile Leu Asp
885 890 895
Leu Lys Asn Ala Ile Ile Asp Ala Lys Ile Val Glu Asn Asp Glu Thr
900 905 910
Tyr Ile Lys Asn Tyr Ile Phe Glu Ser Asn Glu Glu Lys Leu Lys Lys
915 920 925
Arg Leu Phe Cys Glu Glu Leu Val Asp Lys Glu Asp Ile Arg Lys Ile
930 935 940
Phe Glu Asp Glu Asn Phe Lys Phe Lys Ser Phe Ile Lys Lys Asn Glu
945 950 955 960
Ile Gly Asn Phe Lys Ile Asn Phe Gly Ile Leu Ser Asn Leu Glu Cys
965 970 975
Asn Ser Glu Val Glu Ala Lys Lys Ile Ile Gly Lys Asn Ser Lys Lys
980 985 990
Leu Glu Ser Phe Ile Gln Asn Ile Ile Asp Glu Tyr Lys Ser Asn Ile
995 1000 1005
Arg Thr Leu Phe Ser Ser Glu Phe Leu Glu Lys Tyr Lys Glu Glu
1010 1015 1020
Ile Asp Asn Leu Val Glu Asp Thr Glu Ser Glu Asn Lys Asn Lys
1025 1030 1035
Phe Glu Lys Ile Tyr Tyr Pro Lys Glu His Lys Asn Glu Leu Tyr
1040 1045 1050
Ile Tyr Lys Lys Asn Leu Phe Leu Asn Ile Gly Asn Pro Asn Phe
1055 1060 1065
Asp Lys Ile Tyr Gly Leu Ile Ser Lys Asp Ile Lys Asn Val Asp
1070 1075 1080
Thr Lys Ile Leu Phe Asp Asp Asp Ile Lys Lys Asn Lys Ile Ser
1085 1090 1095
Glu Ile Asp Ala Ile Leu Lys Asn Leu Asn Asp Lys Leu Asn Gly
1100 1105 1110
Tyr Ser Asn Asp Tyr Lys Ala Lys Tyr Val Asn Lys Leu Lys Glu
1115 1120 1125
Asn Asp Asp Phe Phe Ala Lys Asn Ile Gln Asn Glu Asn Tyr Ser
1130 1135 1140
Ser Phe Gly Glu Phe Glu Lys Asp Tyr Asn Lys Val Ser Glu Tyr
1145 1150 1155
Lys Lys Ile Arg Asp Leu Val Glu Phe Asn Tyr Leu Asn Lys Ile
1160 1165 1170
Glu Ser Tyr Leu Ile Asp Ile Asn Trp Lys Leu Ala Ile Gln Met
1175 1180 1185
Ala Arg Phe Glu Arg Asp Met His Tyr Ile Val Asn Gly Leu Arg
1190 1195 1200
Glu Leu Gly Ile Ile Lys Leu Ser Gly Tyr Asn Thr Gly Ile Ser
1205 1210 1215
Arg Ala Tyr Pro Lys Arg Asn Gly Ser Asp Gly Phe Tyr Thr Thr
1220 1225 1230
Thr Ala Tyr Tyr Lys Phe Phe Asp Glu Glu Ser Tyr Lys Lys Phe
1235 1240 1245
Glu Lys Ile Cys Tyr Gly Phe Gly Ile Asp Leu Ser Glu Asn Ser
1250 1255 1260
Glu Ile Asn Lys Pro Glu Asn Glu Ser Ile Arg Asn Tyr Ile Ser
1265 1270 1275
His Phe Tyr Ile Val Arg Asn Pro Phe Ala Asp Tyr Ser Ile Ala
1280 1285 1290
Glu Gln Ile Asp Arg Val Ser Asn Leu Leu Ser Tyr Ser Thr Arg
1295 1300 1305
Tyr Asn Asn Ser Thr Tyr Ala Ser Val Phe Glu Val Phe Lys Lys
1310 1315 1320
Asp Val Asn Leu Asp Tyr Asp Glu Leu Lys Lys Lys Phe Arg Leu
1325 1330 1335
Ile Gly Asn Asn Asp Ile Leu Glu Arg Leu Met Lys Pro Lys Lys
1340 1345 1350
Val Ser Val Leu Glu Leu Glu Ser Tyr Asn Ser Asp Tyr Ile Lys
1355 1360 1365
Asn Leu Ile Ile Glu Leu Leu Thr Lys Ile Glu Asn Thr Asn Asp
1370 1375 1380
Thr Leu
1385
<210> 591
<211> 1389
<212> PRT
<213> Leptotrichia shahii
<400> 591
Met Gly Asn Leu Phe Gly His Lys Arg Trp Tyr Glu Val Arg Asp Lys
1 5 10 15
Lys Asp Phe Lys Ile Lys Arg Lys Val Lys Val Lys Arg Asn Tyr Asp
20 25 30
Gly Asn Lys Tyr Ile Leu Asn Ile Asn Glu Asn Asn Asn Lys Glu Lys
35 40 45
Ile Asp Asn Asn Lys Phe Ile Arg Lys Tyr Ile Asn Tyr Lys Lys Asn
50 55 60
Asp Asn Ile Leu Lys Glu Phe Thr Arg Lys Phe His Ala Gly Asn Ile
65 70 75 80
Leu Phe Lys Leu Lys Gly Lys Glu Gly Ile Ile Arg Ile Glu Asn Asn
85 90 95
Asp Asp Phe Leu Glu Thr Glu Glu Val Val Leu Tyr Ile Glu Ala Tyr
100 105 110
Gly Lys Ser Glu Lys Leu Lys Ala Leu Gly Ile Thr Lys Lys Lys Ile
115 120 125
Ile Asp Glu Ala Ile Arg Gln Gly Ile Thr Lys Asp Asp Lys Lys Ile
130 135 140
Glu Ile Lys Arg Gln Glu Asn Glu Glu Glu Ile Glu Ile Asp Ile Arg
145 150 155 160
Asp Glu Tyr Thr Asn Lys Thr Leu Asn Asp Cys Ser Ile Ile Leu Arg
165 170 175
Ile Ile Glu Asn Asp Glu Leu Glu Thr Lys Lys Ser Ile Tyr Glu Ile
180 185 190
Phe Lys Asn Ile Asn Met Ser Leu Tyr Lys Ile Ile Glu Lys Ile Ile
195 200 205
Glu Asn Glu Thr Glu Lys Val Phe Glu Asn Arg Tyr Tyr Glu Glu His
210 215 220
Leu Arg Glu Lys Leu Leu Lys Asp Asp Lys Ile Asp Val Ile Leu Thr
225 230 235 240
Asn Phe Met Glu Ile Arg Glu Lys Ile Lys Ser Asn Leu Glu Ile Leu
245 250 255
Gly Phe Val Lys Phe Tyr Leu Asn Val Gly Gly Asp Lys Lys Lys Ser
260 265 270
Lys Asn Lys Lys Met Leu Val Glu Lys Ile Leu Asn Ile Asn Val Asp
275 280 285
Leu Thr Val Glu Asp Ile Ala Asp Phe Val Ile Lys Glu Leu Glu Phe
290 295 300
Trp Asn Ile Thr Lys Arg Ile Glu Lys Val Lys Lys Val Asn Asn Glu
305 310 315 320
Phe Leu Glu Lys Arg Arg Asn Arg Thr Tyr Ile Lys Ser Tyr Val Leu
325 330 335
Leu Asp Lys His Glu Lys Phe Lys Ile Glu Arg Glu Asn Lys Lys Asp
340 345 350
Lys Ile Val Lys Phe Phe Val Glu Asn Ile Lys Asn Asn Ser Ile Lys
355 360 365
Glu Lys Ile Glu Lys Ile Leu Ala Glu Phe Lys Ile Asp Glu Leu Ile
370 375 380
Lys Lys Leu Glu Lys Glu Leu Lys Lys Gly Asn Cys Asp Thr Glu Ile
385 390 395 400
Phe Gly Ile Phe Lys Lys His Tyr Lys Val Asn Phe Asp Ser Lys Lys
405 410 415
Phe Ser Lys Lys Ser Asp Glu Glu Lys Glu Leu Tyr Lys Ile Ile Tyr
420 425 430
Arg Tyr Leu Lys Gly Arg Ile Glu Lys Ile Leu Val Asn Glu Gln Lys
435 440 445
Val Arg Leu Lys Lys Met Glu Lys Ile Glu Ile Glu Lys Ile Leu Asn
450 455 460
Glu Ser Ile Leu Ser Glu Lys Ile Leu Lys Arg Val Lys Gln Tyr Thr
465 470 475 480
Leu Glu His Ile Met Tyr Leu Gly Lys Leu Arg His Asn Asp Ile Asp
485 490 495
Met Thr Thr Val Asn Thr Asp Asp Phe Ser Arg Leu His Ala Lys Glu
500 505 510
Glu Leu Asp Leu Glu Leu Ile Thr Phe Phe Ala Ser Thr Asn Met Glu
515 520 525
Leu Asn Lys Ile Phe Ser Arg Glu Asn Ile Asn Asn Asp Glu Asn Ile
530 535 540
Asp Phe Phe Gly Gly Asp Arg Glu Lys Asn Tyr Val Leu Asp Lys Lys
545 550 555 560
Ile Leu Asn Ser Lys Ile Lys Ile Ile Arg Asp Leu Asp Phe Ile Asp
565 570 575
Asn Lys Asn Asn Ile Thr Asn Asn Phe Ile Arg Lys Phe Thr Lys Ile
580 585 590
Gly Thr Asn Glu Arg Asn Arg Ile Leu His Ala Ile Ser Lys Glu Arg
595 600 605
Asp Leu Gln Gly Thr Gln Asp Asp Tyr Asn Lys Val Ile Asn Ile Ile
610 615 620
Gln Asn Leu Lys Ile Ser Asp Glu Glu Val Ser Lys Ala Leu Asn Leu
625 630 635 640
Asp Val Val Phe Lys Asp Lys Lys Asn Ile Ile Thr Lys Ile Asn Asp
645 650 655
Ile Lys Ile Ser Glu Glu Asn Asn Asn Asp Ile Lys Tyr Leu Pro Ser
660 665 670
Phe Ser Lys Val Leu Pro Glu Ile Leu Asn Leu Tyr Arg Asn Asn Pro
675 680 685
Lys Asn Glu Pro Phe Asp Thr Ile Glu Thr Glu Lys Ile Val Leu Asn
690 695 700
Ala Leu Ile Tyr Val Asn Lys Glu Leu Tyr Lys Lys Leu Ile Leu Glu
705 710 715 720
Asp Asp Leu Glu Glu Asn Glu Ser Lys Asn Ile Phe Leu Gln Glu Leu
725 730 735
Lys Lys Thr Leu Gly Asn Ile Asp Glu Ile Asp Glu Asn Ile Ile Glu
740 745 750
Asn Tyr Tyr Lys Asn Ala Gln Ile Ser Ala Ser Lys Gly Asn Asn Lys
755 760 765
Ala Ile Lys Lys Tyr Gln Lys Lys Val Ile Glu Cys Tyr Ile Gly Tyr
770 775 780
Leu Arg Lys Asn Tyr Glu Glu Leu Phe Asp Phe Ser Asp Phe Lys Met
785 790 795 800
Asn Ile Gln Glu Ile Lys Lys Gln Ile Lys Asp Ile Asn Asp Asn Lys
805 810 815
Thr Tyr Glu Arg Ile Thr Val Lys Thr Ser Asp Lys Thr Ile Val Ile
820 825 830
Asn Asp Asp Phe Glu Tyr Ile Ile Ser Ile Phe Ala Leu Leu Asn Ser
835 840 845
Asn Ala Val Ile Asn Lys Ile Arg Asn Arg Phe Phe Ala Thr Ser Val
850 855 860
Trp Leu Asn Thr Ser Glu Tyr Gln Asn Ile Ile Asp Ile Leu Asp Glu
865 870 875 880
Ile Met Gln Leu Asn Thr Leu Arg Asn Glu Cys Ile Thr Glu Asn Trp
885 890 895
Asn Leu Asn Leu Glu Glu Phe Ile Gln Lys Met Lys Glu Ile Glu Lys
900 905 910
Asp Phe Asp Asp Phe Lys Ile Gln Thr Lys Lys Glu Ile Phe Asn Asn
915 920 925
Tyr Tyr Glu Asp Ile Lys Asn Asn Ile Leu Thr Glu Phe Lys Asp Asp
930 935 940
Ile Asn Gly Cys Asp Val Leu Glu Lys Lys Leu Glu Lys Ile Val Ile
945 950 955 960
Phe Asp Asp Glu Thr Lys Phe Glu Ile Asp Lys Lys Ser Asn Ile Leu
965 970 975
Gln Asp Glu Gln Arg Lys Leu Ser Asn Ile Asn Lys Lys Asp Leu Lys
980 985 990
Lys Lys Val Asp Gln Tyr Ile Lys Asp Lys Asp Gln Glu Ile Lys Ser
995 1000 1005
Lys Ile Leu Cys Arg Ile Ile Phe Asn Ser Asp Phe Leu Lys Lys
1010 1015 1020
Tyr Lys Lys Glu Ile Asp Asn Leu Ile Glu Asp Met Glu Ser Glu
1025 1030 1035
Asn Glu Asn Lys Phe Gln Glu Ile Tyr Tyr Pro Lys Glu Arg Lys
1040 1045 1050
Asn Glu Leu Tyr Ile Tyr Lys Lys Asn Leu Phe Leu Asn Ile Gly
1055 1060 1065
Asn Pro Asn Phe Asp Lys Ile Tyr Gly Leu Ile Ser Asn Asp Ile
1070 1075 1080
Lys Met Ala Asp Ala Lys Phe Leu Phe Asn Ile Asp Gly Lys Asn
1085 1090 1095
Ile Arg Lys Asn Lys Ile Ser Glu Ile Asp Ala Ile Leu Lys Asn
1100 1105 1110
Leu Asn Asp Lys Leu Asn Gly Tyr Ser Lys Glu Tyr Lys Glu Lys
1115 1120 1125
Tyr Ile Lys Lys Leu Lys Glu Asn Asp Asp Phe Phe Ala Lys Asn
1130 1135 1140
Ile Gln Asn Lys Asn Tyr Lys Ser Phe Glu Lys Asp Tyr Asn Arg
1145 1150 1155
Val Ser Glu Tyr Lys Lys Ile Arg Asp Leu Val Glu Phe Asn Tyr
1160 1165 1170
Leu Asn Lys Ile Glu Ser Tyr Leu Ile Asp Ile Asn Trp Lys Leu
1175 1180 1185
Ala Ile Gln Met Ala Arg Phe Glu Arg Asp Met His Tyr Ile Val
1190 1195 1200
Asn Gly Leu Arg Glu Leu Gly Ile Ile Lys Leu Ser Gly Tyr Asn
1205 1210 1215
Thr Gly Ile Ser Arg Ala Tyr Pro Lys Arg Asn Gly Ser Asp Gly
1220 1225 1230
Phe Tyr Thr Thr Thr Ala Tyr Tyr Lys Phe Phe Asp Glu Glu Ser
1235 1240 1245
Tyr Lys Lys Phe Glu Lys Ile Cys Tyr Gly Phe Gly Ile Asp Leu
1250 1255 1260
Ser Glu Asn Ser Glu Ile Asn Lys Pro Glu Asn Glu Ser Ile Arg
1265 1270 1275
Asn Tyr Ile Ser His Phe Tyr Ile Val Arg Asn Pro Phe Ala Asp
1280 1285 1290
Tyr Ser Ile Ala Glu Gln Ile Asp Arg Val Ser Asn Leu Leu Ser
1295 1300 1305
Tyr Ser Thr Arg Tyr Asn Asn Ser Thr Tyr Ala Ser Val Phe Glu
1310 1315 1320
Val Phe Lys Lys Asp Val Asn Leu Asp Tyr Asp Glu Leu Lys Lys
1325 1330 1335
Lys Phe Lys Leu Ile Gly Asn Asn Asp Ile Leu Glu Arg Leu Met
1340 1345 1350
Lys Pro Lys Lys Val Ser Val Leu Glu Leu Glu Ser Tyr Asn Ser
1355 1360 1365
Asp Tyr Ile Lys Asn Leu Ile Ile Glu Leu Leu Thr Lys Ile Glu
1370 1375 1380
Asn Thr Asn Asp Thr Leu
1385
<210> 592
<211> 10
<212> PRT
<213> Lactococcus lactis
<400> 592
Phe Leu Val Asn His Asn Tyr Tyr Ser Phe
1 5 10
<210> 593
<211> 16
<212> PRT
<213> Lactococcus lactis
<400> 593
Leu Gln Lys Phe Thr Gly Asp Ile Glu Asn Leu Val Lys Ala Ser Leu
1 5 10 15
<210> 594
<211> 9
<212> PRT
<213> Lactococcus lactis
<400> 594
Val Ile Val Pro Glu Leu Thr Phe Gly
1 5
<210> 595
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 595
Trp Ile Arg Ala Gly Trp Phe Ile Arg Asn Arg Ser Ala His Tyr
1 5 10 15
<210> 596
<211> 13
<212> PRT
<213> Lactococcus lactis
<400> 596
Asn Lys Asp Leu Phe Ala Phe Met Leu Ser Ile Lys Gln
1 5 10
<210> 597
<211> 10
<212> PRT
<213> Lactococcus lactis
<400> 597
Phe Leu His Lys Asn Ser Tyr Phe Arg Phe
1 5 10
<210> 598
<211> 16
<212> PRT
<213> Lactococcus lactis
<400> 598
Leu Phe Ile Phe Ser Thr Arg Leu Glu Ile Phe Trp Lys Lys Lys Ile
1 5 10 15
<210> 599
<211> 9
<212> PRT
<213> Lactococcus lactis
<400> 599
Ala Leu Val Glu Glu Leu Thr Phe Gly
1 5
<210> 600
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 600
Trp Met Asn Val Val Arg Leu Tyr Arg Asn Lys Ser Ala His Gly
1 5 10 15
<210> 601
<211> 13
<212> PRT
<213> Lactococcus lactis
<400> 601
Lys Ser Tyr Leu Tyr Gly Ala Leu Tyr Val Phe Lys His
1 5 10
<210> 602
<211> 10
<212> PRT
<213> Corynebacterium diphtheriae
<400> 602
Leu Leu Ala Gln Leu Asn Tyr Tyr Arg Leu
1 5 10
<210> 603
<211> 16
<212> PRT
<213> Corynebacterium diphtheriae
<400> 603
Val Phe Ile Glu Leu Asp Arg Val Glu Leu Ala Ile Gln Thr Arg Leu
1 5 10 15
<210> 604
<211> 9
<212> PRT
<213> Corynebacterium diphtheriae
<400> 604
Ala Ala Val Glu Val Met Asp Trp Gly
1 5
<210> 605
<211> 15
<212> PRT
<213> Corynebacterium diphtheriae
<400> 605
Trp Leu Lys Ser Leu Asn Ile Leu Arg Asn Tyr Ala Ala His His
1 5 10 15
<210> 606
<211> 13
<212> PRT
<213> Corynebacterium diphtheriae
<400> 606
Gly Gln Leu Ser Met Ile Gln Tyr Leu His His Gln Leu
1 5 10
<210> 607
<211> 12
<212> PRT
<213> Shewanella baltica
<400> 607
Met Leu Ile Glu Asn Asp Leu Asp Gly Ile Glu Asn
1 5 10
<210> 608
<211> 16
<212> PRT
<213> Shewanella baltica
<400> 608
Asn Tyr Gln Leu Phe Tyr Phe Leu Glu Lys Thr Ile Arg Asn Gln Ile
1 5 10 15
<210> 609
<211> 15
<212> PRT
<213> Shewanella baltica
<400> 609
Val Met Phe Asn Leu Asn Thr Leu Arg Asn Pro Ile Ala His Cys
1 5 10 15
<210> 610
<211> 13
<212> PRT
<213> Shewanella baltica
<400> 610
Asp Glu Lys Leu Arg Leu Glu Ile Ser Leu Arg Asp Trp
1 5 10
<210> 611
<211> 12
<212> PRT
<213> Lactococcus lactis
<400> 611
Leu Arg Glu Ile Asn Ile Lys Ala Ser Lys Ser Arg
1 5 10
<210> 612
<211> 16
<212> PRT
<213> Lactococcus lactis
<400> 612
Leu Leu Pro Leu Leu His Lys Tyr Glu Trp Ser Leu Arg Lys Leu Ile
1 5 10 15
<210> 613
<211> 9
<212> PRT
<213> Lactococcus lactis
<400> 613
Tyr Asp Phe Glu Glu Tyr Leu Phe Gly
1 5
<210> 614
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 614
Asp Met Arg Leu Ile Arg Asp Gly Arg Asn Ile Val Gly His Asn
1 5 10 15
<210> 615
<211> 13
<212> PRT
<213> Lactococcus lactis
<400> 615
Leu Ser Lys Gly Leu Lys Lys Tyr Ile Lys Lys Leu Asp
1 5 10
<210> 616
<211> 12
<212> PRT
<213> Geobacter bemidjiensis
<400> 616
Arg Leu Pro Leu Thr Ser His Ile Gln Lys Gln Asp
1 5 10
<210> 617
<211> 16
<212> PRT
<213> Geobacter bemidjiensis
<400> 617
Ile Tyr Pro Lys Leu Asn Arg Ile Glu Asn Arg Leu Arg His Tyr Leu
1 5 10 15
<210> 618
<211> 9
<212> PRT
<213> Geobacter bemidjiensis
<400> 618
Phe Glu Leu Gly Lys Ile Val Tyr Ala
1 5
<210> 619
<211> 15
<212> PRT
<213> Geobacter bemidjiensis
<400> 619
Lys Trp Ile Arg Leu Glu Glu Ile Arg His Lys Val Ala His Asn
1 5 10 15
<210> 620
<211> 13
<212> PRT
<213> Geobacter bemidjiensis
<400> 620
Ala Asn Glu Tyr Ile Asp Ser Leu Gln Ser Ile Ile Asp
1 5 10
<210> 621
<211> 12
<212> PRT
<213> Salmonella enterica
<400> 621
Phe Val Thr Ser Leu Glu His Leu Arg Gln Gln Gln
1 5 10
<210> 622
<211> 16
<212> PRT
<213> Salmonella enterica
<400> 622
Ala Gln Arg Gln Leu Arg Ala Ile Glu Leu Thr Leu Lys Ala Leu Ile
1 5 10 15
<210> 623
<211> 9
<212> PRT
<213> Salmonella enterica
<400> 623
Asn His Tyr Leu Lys Gln His Phe Gly
1 5
<210> 624
<211> 15
<212> PRT
<213> Salmonella enterica
<400> 624
Phe Leu Asp Asp Cys Arg Leu Ala Arg Asn Glu Val Ile Ala Arg
1 5 10 15
<210> 625
<211> 13
<212> PRT
<213> Salmonella enterica
<400> 625
Leu Met Leu Leu Asn Val Gln Tyr Gln Gln Ile Val Arg
1 5 10
<210> 626
<211> 12
<212> PRT
<213> Shigella flexneri
<400> 626
Phe Leu Trp Gln Leu Glu Tyr Leu Arg Glu Lys Gln
1 5 10
<210> 627
<211> 16
<212> PRT
<213> Shigella flexneri
<400> 627
Ser Leu Gln Gln Val Arg Ala Leu Glu Leu Thr Ile Arg Ser Leu Ile
1 5 10 15
<210> 628
<211> 9
<212> PRT
<213> Shigella flexneri
<400> 628
Leu Glu His Leu Asn Lys Leu Phe Gly
1 5
<210> 629
<211> 15
<212> PRT
<213> Shigella flexneri
<400> 629
Phe Leu Asp Asp Ile Arg Val Ile Arg Asn Arg Leu Ala His His
1 5 10 15
<210> 630
<211> 13
<212> PRT
<213> Shigella flexneri
<400> 630
Thr Thr Leu Val Asn Tyr Tyr Tyr Arg Glu Ile Thr Glu
1 5 10
<210> 631
<211> 16
<212> PRT
<213> Streptomyces avermitilis
<400> 631
Ala Tyr Ile Trp Leu Asn Leu Val Glu Gln Arg Leu Arg Ala Val Val
1 5 10 15
<210> 632
<211> 9
<212> PRT
<213> Streptomyces avermitilis
<400> 632
Asn Val Leu Ser Phe Leu Thr Leu Pro
1 5
<210> 633
<211> 11
<212> PRT
<213> Streptomyces avermitilis
<400> 633
Leu Glu Val Thr Arg Asn Val Val Ser Arg Asn
1 5 10
<210> 634
<211> 13
<212> PRT
<213> Streptomyces avermitilis
<400> 634
Arg Tyr Gly Asp Val Val Gly Val His Pro Asp Arg Val
1 5 10
<210> 635
<211> 10
<212> PRT
<213> Helicobacter pylori
<400> 635
Ser Ile Ser Val Leu His Tyr Asp Tyr Leu
1 5 10
<210> 636
<211> 16
<212> PRT
<213> Helicobacter pylori
<400> 636
Leu Phe Leu Trp Ile His Phe Phe Glu Thr Ala Leu Arg Ser Lys Met
1 5 10 15
<210> 637
<211> 9
<212> PRT
<213> Helicobacter pylori
<400> 637
Gln Ile Leu Asn Leu Phe Thr Leu Gly
1 5
<210> 638
<211> 15
<212> PRT
<213> Helicobacter pylori
<400> 638
Thr Phe Ser Leu Ile Arg Lys Ala Arg Asn Asp Leu Phe His Asn
1 5 10 15
<210> 639
<211> 13
<212> PRT
<213> Helicobacter pylori
<400> 639
Thr Leu Lys Leu Glu Arg Ala Ile Phe Phe Lys Thr Ile
1 5 10
<210> 640
<211> 11
<212> PRT
<213> Methylomirabilis oxyfera
<400> 640
Gly Pro Pro Glu Tyr Tyr Tyr Arg Leu Cys Arg
1 5 10
<210> 641
<211> 16
<212> PRT
<213> Methylomirabilis oxyfera
<400> 641
Ala Asp Ser Lys Leu Lys Asp Thr Val Ser Glu Met Arg Lys Phe Ile
1 5 10 15
<210> 642
<211> 15
<212> PRT
<213> Methylomirabilis oxyfera
<400> 642
Trp Met Asn Arg Ile Asn Glu Leu Arg Arg Ile Pro Ala His Pro
1 5 10 15
<210> 643
<211> 13
<212> PRT
<213> Methylomirabilis oxyfera
<400> 643
Asp Phe Glu Tyr Ile Asp Phe Ile Tyr Asp Glu Leu Met
1 5 10
<210> 644
<211> 12
<212> PRT
<213> Novosphingobium aromaticivorans
<400> 644
Thr Ala Val Lys Gln Gln Ser Phe Gly Met Glu Ala
1 5 10
<210> 645
<211> 16
<212> PRT
<213> Novosphingobium aromaticivorans
<400> 645
Ala Ala Ala Lys Val Thr Gln Ile His Lys Lys Leu Phe Asn Tyr Val
1 5 10 15
<210> 646
<211> 15
<212> PRT
<213> Novosphingobium aromaticivorans
<400> 646
Trp Ile Lys Val Leu Asn Asp Ile Arg Gln Tyr Thr Ala His Pro
1 5 10 15
<210> 647
<211> 13
<212> PRT
<213> Novosphingobium aromaticivorans
<400> 647
Gln Val Ser Phe Val Asn Glu Val Tyr Glu Lys Val Glu
1 5 10
<210> 648
<211> 11
<212> PRT
<213> Elizabethkingia anophelis
<400> 648
Gly Glu Ile Lys Tyr Trp Arg Thr Phe Gln Lys
1 5 10
<210> 649
<211> 16
<212> PRT
<213> Elizabethkingia anophelis
<400> 649
Ala Ile Ala Tyr Ile Arg Asp Ile Glu Thr Glu Phe Lys Ser Asp Phe
1 5 10 15
<210> 650
<211> 15
<212> PRT
<213> Elizabethkingia anophelis
<400> 650
Trp Met Val Lys Leu Glu Arg Ile Arg Asn Gln Asn Phe His Ser
1 5 10 15
<210> 651
<211> 13
<212> PRT
<213> Elizabethkingia anophelis
<400> 651
Glu Leu Ser Phe Leu Glu Glu Leu His Asp Trp Ile Tyr
1 5 10
<210> 652
<211> 12
<212> PRT
<213> Escherichia coli
<400> 652
Phe Ser Ala Leu Pro Arg Ile Ile Glu Tyr Ala Tyr
1 5 10
<210> 653
<211> 16
<212> PRT
<213> Escherichia coli
<400> 653
Pro Phe Leu Leu Leu Ser Glu Ile Glu Asn His Ile Arg Lys Leu Ile
1 5 10 15
<210> 654
<211> 9
<212> PRT
<213> Escherichia coli
<400> 654
Glu Ser Val Ala Asp Leu Thr Phe Gly
1 5
<210> 655
<211> 15
<212> PRT
<213> Escherichia coli
<400> 655
Glu Leu Asp Lys Val Arg Ile Ile Arg Asn Asp Val Met His Phe
1 5 10 15
<210> 656
<211> 13
<212> PRT
<213> Escherichia coli
<400> 656
Asn His Glu Leu Leu His Asn Phe Val Arg Phe Ile His
1 5 10
<210> 657
<211> 12
<212> PRT
<213> Haloarcula marismortui
<400> 657
Phe Glu Leu Phe Asp Thr Leu Ala Glu Asp Asp Tyr
1 5 10
<210> 658
<211> 16
<212> PRT
<213> Haloarcula marismortui
<400> 658
Pro Phe Leu Gln Ile Gly Glu Ile Glu Glu Ser Leu Arg His Leu Phe
1 5 10 15
<210> 659
<211> 9
<212> PRT
<213> Haloarcula marismortui
<400> 659
Asp Arg Pro Glu Asp Phe Ser Phe Asp
1 5
<210> 660
<211> 15
<212> PRT
<213> Haloarcula marismortui
<400> 660
Leu Leu Glu Asp Ile Arg Glu Thr Arg Asn Ala Leu Leu His Phe
1 5 10 15
<210> 661
<211> 13
<212> PRT
<213> Haloarcula marismortui
<400> 661
Asp Arg Asp Gln Leu Asp Met Ala His Gly Tyr Phe Thr
1 5 10
<210> 662
<211> 12
<212> PRT
<213> Nostoc sp.
<400> 662
Met Lys Leu Leu Pro Ile Leu Gln Gln Asn Pro Arg
1 5 10
<210> 663
<211> 15
<212> PRT
<213> Nostoc sp.
<400> 663
Phe Gly Leu Val Thr Leu Leu Glu Met Asn Leu Leu Arg Leu Val
1 5 10 15
<210> 664
<211> 9
<212> PRT
<213> Nostoc sp.
<400> 664
Asp Leu Leu Asp Tyr Leu Gln Phe Cys
1 5
<210> 665
<211> 15
<212> PRT
<213> Nostoc sp.
<400> 665
Phe Leu Lys Ser Ala Glu Gln Leu Arg Asn Arg Leu Ala His Ala
1 5 10 15
<210> 666
<211> 13
<212> PRT
<213> Nostoc sp.
<400> 666
Ser Trp Asn Asp Leu Ile Ser Leu Ala Glu Ala Met Glu
1 5 10
<210> 667
<211> 10
<212> PRT
<213> Xanthobacter autotrophicus
<400> 667
Val Phe Glu Gly Met Glu Leu Leu Pro Ala
1 5 10
<210> 668
<211> 14
<212> PRT
<213> Xanthobacter autotrophicus
<400> 668
Ala Leu Ile Pro Phe Val Glu Lys Arg Leu Glu Thr Ser Leu
1 5 10
<210> 669
<211> 8
<212> PRT
<213> Xanthobacter autotrophicus
<400> 669
Glu Ala Phe Lys Ala Val Leu Gly
1 5
<210> 670
<211> 15
<212> PRT
<213> Xanthobacter autotrophicus
<400> 670
Leu Val Asn Glu Leu Gly Asp Val Arg Asn Lys Leu Ser His Asn
1 5 10 15
<210> 671
<211> 13
<212> PRT
<213> Xanthobacter autotrophicus
<400> 671
Tyr Asp Asp Ala Glu Arg Ala Leu Asp Thr Met Arg Arg
1 5 10
<210> 672
<211> 10
<212> PRT
<213> Methanospirillum hungatei
<400> 672
Val Gly Arg Ala Met Asp Gln Leu Lys Thr
1 5 10
<210> 673
<211> 14
<212> PRT
<213> Methanospirillum hungatei
<400> 673
Gly Leu Met Arg Phe Val Glu Arg Glu Met Lys Ser Ala Tyr
1 5 10
<210> 674
<211> 8
<212> PRT
<213> Methanospirillum hungatei
<400> 674
Lys Val Phe Ser Gln Ile Leu Gly
1 5
<210> 675
<211> 15
<212> PRT
<213> Methanospirillum hungatei
<400> 675
Leu Val Ser Glu Leu Arg Glu Thr Arg Asn Gln Trp Ala His Gln
1 5 10 15
<210> 676
<211> 13
<212> PRT
<213> Methanospirillum hungatei
<400> 676
Thr Asn Asp Thr Leu Arg Ala Leu Asp Ser Thr Ala Arg
1 5 10
<210> 677
<211> 10
<212> PRT
<213> Roseiflexus sp.
<400> 677
Ile Gly Lys Ala Leu Asp Leu Leu Arg Gln
1 5 10
<210> 678
<211> 14
<212> PRT
<213> Roseiflexus sp.
<400> 678
Gly Leu Gln Pro Phe Ile Glu Arg Glu Leu Gln Asn His Tyr
1 5 10
<210> 679
<211> 8
<212> PRT
<213> Roseiflexus sp.
<400> 679
Asp Val Phe Arg Lys Thr Leu Gly
1 5
<210> 680
<211> 15
<212> PRT
<213> Roseiflexus sp.
<400> 680
Leu Val Ser Glu Leu Arg Glu Trp Arg Asn Lys Trp Ala His Gln
1 5 10 15
<210> 681
<211> 13
<212> PRT
<213> Roseiflexus sp.
<400> 681
Thr Asp Asp Thr Tyr Arg Val Leu Asp Ser Ala Ala Arg
1 5 10
<210> 682
<211> 10
<212> PRT
<213> Plasmodium yoelii
<400> 682
Ile Leu Asn Ile Phe His Ile Leu Ser Ala
1 5 10
<210> 683
<211> 14
<212> PRT
<213> Plasmodium yoelii
<400> 683
His Leu Ser Pro Ile Ile Glu Gln Ile Met Glu Met Glu Tyr
1 5 10
<210> 684
<211> 7
<212> PRT
<213> Plasmodium yoelii
<400> 684
Asp Ile Phe Glu Asn Arg Ile
1 5
<210> 685
<211> 15
<212> PRT
<213> Plasmodium yoelii
<400> 685
Ile Leu Glu Asn Leu Gln Lys Ala Ser Ile Phe Trp Ala Asn Gln
1 5 10 15
<210> 686
<211> 13
<212> PRT
<213> Plasmodium yoelii
<400> 686
Glu Phe Phe Leu Ser Asn Leu Val Ser Ser Tyr Phe Phe
1 5 10
<210> 687
<211> 10
<212> PRT
<213> Theileria parva
<400> 687
Val Val Met Ile Phe Gln Cys Val Cys Asp
1 5 10
<210> 688
<211> 14
<212> PRT
<213> Theileria parva
<400> 688
Ala Phe Gln Pro Phe Ile Ser Lys Cys Met Leu Lys Lys Phe
1 5 10
<210> 689
<211> 7
<212> PRT
<213> Theileria parva
<400> 689
Asp Ile Phe Glu Gln Val Leu
1 5
<210> 690
<211> 15
<212> PRT
<213> Theileria parva
<400> 690
His Leu Asn Thr Ile Gln Thr Ala Ser Ile Tyr Trp Ala Asn Gln
1 5 10 15
<210> 691
<211> 8
<212> PRT
<213> Theileria parva
<400> 691
Asn Tyr Gly Lys Cys Arg Lys Ile
1 5
<210> 692
<211> 10
<212> PRT
<213> Daphnia pulex
<400> 692
Ser Ser Lys Glu Ser Ala Ala Ile Ala Ile
1 5 10
<210> 693
<211> 14
<212> PRT
<213> Daphnia pulex
<400> 693
Gly His Ile Val Phe Asp Thr Phe Leu Glu Asp Val Ala Pro
1 5 10
<210> 694
<211> 8
<212> PRT
<213> Daphnia pulex
<400> 694
Asp Cys Phe Ile Ile Pro Pro Gly
1 5
<210> 695
<211> 15
<212> PRT
<213> Daphnia pulex
<400> 695
Ile Leu Glu Arg Ala Met Asp Gly Arg His Ala Val Ser His His
1 5 10 15
<210> 696
<211> 13
<212> PRT
<213> Daphnia pulex
<400> 696
Trp Glu Gln His Leu Lys Asp Tyr Val Tyr Ile Leu Thr
1 5 10
<210> 697
<211> 10
<212> PRT
<213> Homo sapiens
<400> 697
Ala Gly His Cys Leu Leu Leu Leu Arg Ser
1 5 10
<210> 698
<211> 16
<212> PRT
<213> Homo sapiens
<400> 698
Cys Leu Gln Gly Phe Val Gly Arg Glu Val Leu Ser Phe His Arg Gly
1 5 10 15
<210> 699
<211> 15
<212> PRT
<213> Homo sapiens
<400> 699
Lys Val Thr Glu Val Ile Lys Cys Arg Asn Glu Ile Met His Ser
1 5 10 15
<210> 700
<211> 13
<212> PRT
<213> Homo sapiens
<400> 700
Ser Ser Thr Trp Leu Arg Asp Phe Gln Met Lys Ile Gln
1 5 10
<210> 701
<211> 10
<212> PRT
<213> Branchiostoma floridae
<400> 701
Val Gly Ile Ala Leu Leu Thr Thr Arg Asp
1 5 10
<210> 702
<211> 16
<212> PRT
<213> Branchiostoma floridae
<400> 702
Gly Leu Thr Asn Val Thr Glu Gln Ala Ala Lys Glu Leu Gln Ala Glu
1 5 10 15
<210> 703
<211> 15
<212> PRT
<213> Branchiostoma floridae
<400> 703
Pro Leu Lys Asn Val Ile Glu Val Arg Asn Lys Thr Met His Ser
1 5 10 15
<210> 704
<211> 13
<212> PRT
<213> Branchiostoma floridae
<400> 704
Asp Arg Gln Thr Phe Asn Glu Tyr Met Asp Lys Met Glu
1 5 10
<210> 705
<211> 10
<212> PRT
<213> Homo sapiens
<400> 705
Val Ser Asp Leu Glu Lys Ser Leu Gly Thr
1 5 10
<210> 706
<211> 14
<212> PRT
<213> Homo sapiens
<400> 706
Gly Leu Ser Ser Ile Leu Glu Thr Glu Met Lys Ile Ala Phe
1 5 10
<210> 707
<211> 8
<212> PRT
<213> Homo sapiens
<400> 707
Lys His Trp Leu Ala Val Phe Gly
1 5
<210> 708
<211> 16
<212> PRT
<213> Homo sapiens
<400> 708
Thr Ile Glu Ser Leu Tyr Lys Asn Leu Arg Lys Ala Asn Lys Ala Val
1 5 10 15
<210> 709
<211> 13
<212> PRT
<213> Homo sapiens
<400> 709
Ser Arg Ser Leu Leu His Ala Phe Ser Thr Arg Ser Asn
1 5 10
<210> 710
<211> 10
<212> PRT
<213> Ostreococcus lucimarinus
<400> 710
Met Glu Arg Leu Met Met Val Leu Asp His
1 5 10
<210> 711
<211> 14
<212> PRT
<213> Ostreococcus lucimarinus
<400> 711
Val Leu Ala Ile Val Leu Glu Gly Gly Leu Arg Ala Glu Phe
1 5 10
<210> 712
<211> 8
<212> PRT
<213> Ostreococcus lucimarinus
<400> 712
Ala Asn Trp Gly Ser Leu Phe Ser
1 5
<210> 713
<211> 14
<212> PRT
<213> Ostreococcus lucimarinus
<400> 713
Glu Ile Glu Val Leu Leu Asp Ala Ala Ile Arg Gln Arg Lys
1 5 10
<210> 714
<211> 13
<212> PRT
<213> Ostreococcus lucimarinus
<400> 714
Ala Arg Asp Val Ser Ser Ala Ala Val Ala Leu Leu Asn
1 5 10
<210> 715
<211> 10
<212> PRT
<213> Branchiostoma floridae
<400> 715
Leu Cys Gly Met Lys Thr Leu Leu Lys Ala
1 5 10
<210> 716
<211> 14
<212> PRT
<213> Branchiostoma floridae
<400> 716
Val Leu Ala Val Val Leu Glu Thr Glu Met Lys Ala Val Phe
1 5 10
<210> 717
<211> 8
<212> PRT
<213> Branchiostoma floridae
<400> 717
Lys His Trp Ile Ala Val Phe Gly
1 5
<210> 718
<211> 17
<212> PRT
<213> Branchiostoma floridae
<400> 718
His Leu Asp Ser Leu Val Lys His Phe Thr Arg Gly Arg Ser Tyr Gly
1 5 10 15
Val
<210> 719
<211> 13
<212> PRT
<213> Branchiostoma floridae
<400> 719
Ala Leu Gln Leu Val Arg Gln Leu His Asn His Ser Thr
1 5 10
<210> 720
<211> 10
<212> PRT
<213> Microcystis aeruginosa
<400> 720
Leu Asn Trp Leu Asp Gln Leu His Asp Asp
1 5 10
<210> 721
<211> 16
<212> PRT
<213> Microcystis aeruginosa
<400> 721
Leu Ile Glu Leu Cys Gly Trp Ile Glu Glu Thr Met Asp Asp Ile Val
1 5 10 15
<210> 722
<211> 9
<212> PRT
<213> Microcystis aeruginosa
<400> 722
Phe Arg Lys Met Leu Met Met Val Ile
1 5
<210> 723
<211> 15
<212> PRT
<213> Microcystis aeruginosa
<400> 723
Tyr Leu Gly Asn Leu Lys Asp Ser Arg Asn Arg Ala Ala His Thr
1 5 10 15
<210> 724
<211> 13
<212> PRT
<213> Microcystis aeruginosa
<400> 724
Phe Asp Lys Ile Tyr Gly Leu Leu Lys Glu Leu Asp Ala
1 5 10
<210> 725
<211> 12
<212> PRT
<213> Lactococcus lactis
<400> 725
Leu Ser Glu Leu His Glu Phe Ile Lys Lys Leu Asn
1 5 10
<210> 726
<211> 16
<212> PRT
<213> Lactococcus lactis
<400> 726
Val Ile Arg Ser Cys Gly Ile Ile Glu Gln Leu Thr Lys Thr Leu Ile
1 5 10 15
<210> 727
<211> 9
<212> PRT
<213> Lactococcus lactis
<400> 727
Ile Asn Gly Leu Ile Asp Thr Phe Asp
1 5
<210> 728
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 728
His Ile Asp Ser Leu Arg Gln Leu Arg Asn Ser Ile Ala His Gly
1 5 10 15
<210> 729
<211> 13
<212> PRT
<213> Lactococcus lactis
<400> 729
Met Gly Tyr Phe Asp Ser Cys Ile Ile Leu Met Phe Arg
1 5 10
<210> 730
<211> 12
<212> PRT
<213> Frankia sp.
<400> 730
Leu Ser Glu Leu Ala Ala Leu Val Gln Asp Gln Ala
1 5 10
<210> 731
<211> 16
<212> PRT
<213> Frankia sp.
<400> 731
Val Ile Arg Ser Cys Gly Tyr Leu Glu Gln Thr Val Ala Gly Thr Phe
1 5 10 15
<210> 732
<211> 9
<212> PRT
<213> Frankia sp.
<400> 732
Leu Glu Thr Leu Ala Gly Arg Phe Asp
1 5
<210> 733
<211> 15
<212> PRT
<213> Frankia sp.
<400> 733
Glu Leu Ala Thr Leu Val Asp Arg Arg Asn Arg Ile Ala His Gly
1 5 10 15
<210> 734
<211> 13
<212> PRT
<213> Frankia sp.
<400> 734
Leu Glu Leu His Arg Val Ala Cys Glu Ala Ala Asp Trp
1 5 10
<210> 735
<211> 10
<212> PRT
<213> Neisseria meningitidis
<400> 735
Cys Cys Ser Ile Phe Ser Asp Phe Arg Met
1 5 10
<210> 736
<211> 16
<212> PRT
<213> Neisseria meningitidis
<400> 736
Leu Phe His Val Val Ser Ile Phe Glu Ile Val Leu Arg Asn Lys Ile
1 5 10 15
<210> 737
<211> 9
<212> PRT
<213> Neisseria meningitidis
<400> 737
Gln Leu Val Ala Gly Leu Gly Phe Gly
1 5
<210> 738
<211> 15
<212> PRT
<213> Neisseria meningitidis
<400> 738
Glu Leu Ser Asn Ile Asn Lys Phe Arg Asn Arg Leu Ala His His
1 5 10 15
<210> 739
<211> 13
<212> PRT
<213> Neisseria meningitidis
<400> 739
Asp Val Asp Thr Ala Ser Val Phe Ser His Phe Ser Asp
1 5 10
<210> 740
<211> 10
<212> PRT
<213> Pseudomonas syringae
<400> 740
Leu Glu Lys His Phe Ser Ser Ala Arg Leu
1 5 10
<210> 741
<211> 16
<212> PRT
<213> Pseudomonas syringae
<400> 741
Met Met Pro Met Leu Ser Val Leu Glu Ile Ala Leu Lys Asn Gly Ile
1 5 10 15
<210> 742
<211> 9
<212> PRT
<213> Pseudomonas syringae
<400> 742
Lys Ile Val Ala Glu Leu Ala Phe Gly
1 5
<210> 743
<211> 15
<212> PRT
<213> Pseudomonas syringae
<400> 743
Ala Leu Asn Leu Ile Arg Asn Leu Arg Asn Arg Val Phe His His
1 5 10 15
<210> 744
<211> 13
<212> PRT
<213> Pseudomonas syringae
<400> 744
Asp Pro Gln Leu Val Pro Trp Leu Ala Gln Tyr Asp Arg
1 5 10
<210> 745
<211> 10
<212> PRT
<213> Geobacter uraniireducens
<400> 745
Leu Arg Arg Ala Ile Ser His Glu Arg Leu
1 5 10
<210> 746
<211> 16
<212> PRT
<213> Geobacter uraniireducens
<400> 746
Leu Tyr Thr Pro Leu Gln Cys Leu Glu Val Cys Leu Arg Asn Ser Ile
1 5 10 15
<210> 747
<211> 9
<212> PRT
<213> Geobacter uraniireducens
<400> 747
Arg Ile Ile Pro Glu Leu Thr Phe Gly
1 5
<210> 748
<211> 15
<212> PRT
<213> Geobacter uraniireducens
<400> 748
Arg Phe Asn His Ile Arg Thr Leu Arg Asn Arg Ile Phe His His
1 5 10 15
<210> 749
<211> 13
<212> PRT
<213> Geobacter uraniireducens
<400> 749
Asn Pro Ala Met Met Thr Phe Val Glu Pro Phe Asp Ser
1 5 10
<210> 750
<211> 16
<212> PRT
<213> Sulfuricurvum kujiense
<400> 750
Glu Glu Lys Ser Glu Phe Ile Arg Glu Phe Phe Lys Arg Thr Leu His
1 5 10 15
<210> 751
<211> 9
<212> PRT
<213> Sulfuricurvum kujiense
<400> 751
Thr Gln Thr Ile Asn Ser Phe Leu Gly
1 5
<210> 752
<211> 14
<212> PRT
<213> Sulfuricurvum kujiense
<400> 752
Phe Arg Asn Tyr Leu Lys Arg Leu Arg Asn Ala Val Ser His
1 5 10
<210> 753
<211> 13
<212> PRT
<213> Sulfuricurvum kujiense
<400> 753
Val Asn Leu Leu Ile Thr Leu Leu Ser Arg Asn Ile Leu
1 5 10
<210> 754
<211> 13
<212> PRT
<213> Dethiobacter alkaliphilus
<400> 754
Gln Val Val Glu Lys Asp Phe Val Ala Arg Thr Met His
1 5 10
<210> 755
<211> 9
<212> PRT
<213> Dethiobacter alkaliphilus
<400> 755
Thr Leu Leu Ile Asn Cys Leu Leu Gly
1 5
<210> 756
<211> 14
<212> PRT
<213> Dethiobacter alkaliphilus
<400> 756
Ala Ser Arg Phe Leu Gln Cys Met Arg Asn Ser Val Ala His
1 5 10
<210> 757
<211> 10
<212> PRT
<213> Dethiobacter alkaliphilus
<400> 757
Leu Ala Thr Lys Leu Ala Gln Tyr Val Gln
1 5 10
<210> 758
<211> 13
<212> PRT
<213> Klebsiella pneumoniae
<400> 758
Ser Asp Phe Glu Thr Asp Phe Val Gln Arg Thr Leu Ala
1 5 10
<210> 759
<211> 9
<212> PRT
<213> Klebsiella pneumoniae
<400> 759
Thr Leu Thr Leu Asn Cys Leu Leu Gly
1 5
<210> 760
<211> 14
<212> PRT
<213> Klebsiella pneumoniae
<400> 760
Leu Arg Gln Leu Ile His Lys Met Arg Asn Ser Val Ala His
1 5 10
<210> 761
<211> 13
<212> PRT
<213> Klebsiella pneumoniae
<400> 761
Leu Leu Pro Phe Leu Lys Tyr Tyr Ala Thr Leu Leu Leu
1 5 10
<210> 762
<211> 10
<212> PRT
<213> Lactobacillus casei
<400> 762
Lys Ile Asp Arg Glu Met Phe Trp Arg Arg
1 5 10
<210> 763
<211> 16
<212> PRT
<213> Lactobacillus casei
<400> 763
Tyr Leu Leu Leu Tyr Ser Ser Trp Glu Gly Phe Ile Arg Ser Ile Ala
1 5 10 15
<210> 764
<211> 9
<212> PRT
<213> Lactobacillus casei
<400> 764
Leu Ala Arg Ile Val Ser Val Leu Asp
1 5
<210> 765
<211> 14
<212> PRT
<213> Lactobacillus casei
<400> 765
Asp Arg Asp Leu Leu Lys Val Arg Asn Glu Ile Ala His Gly
1 5 10
<210> 766
<211> 13
<212> PRT
<213> Lactobacillus casei
<400> 766
Thr Val Ser His Val Leu Glu Met Met Asp Leu Phe Ser
1 5 10
<210> 767
<211> 10
<212> PRT
<213> Caulobacter sp.
<400> 767
Asp Leu Asp Ala Ala Arg Leu Arg Arg Ala
1 5 10
<210> 768
<211> 16
<212> PRT
<213> Caulobacter sp.
<400> 768
Ile Val Leu Ala Tyr Ser His Trp Glu Gly Phe Tyr Asn Glu Cys Ile
1 5 10 15
<210> 769
<211> 9
<212> PRT
<213> Caulobacter sp.
<400> 769
Leu Lys Glu Asn Phe Arg Ile Leu Gly
1 5
<210> 770
<211> 14
<212> PRT
<213> Caulobacter sp.
<400> 770
Asn Lys Glu Leu Val Gly Trp Arg His Ser Ile Ala His Gly
1 5 10
<210> 771
<211> 13
<212> PRT
<213> Caulobacter sp.
<400> 771
His Ile Ile Leu Thr Asn Ser Leu Leu Leu Thr Leu Ser
1 5 10
<210> 772
<211> 10
<212> PRT
<213> Microcystis aeruginosa
<400> 772
Asn Leu Asp Glu Asp Met Ala Trp Arg Ile
1 5 10
<210> 773
<211> 16
<212> PRT
<213> Microcystis aeruginosa
<400> 773
Ile Thr Thr Leu Tyr Ala His Trp Glu Gly Phe Ile Lys Tyr Ala Ala
1 5 10 15
<210> 774
<211> 9
<212> PRT
<213> Microcystis aeruginosa
<400> 774
Phe Thr Asp Ile Cys Thr Ile Leu Gly
1 5
<210> 775
<211> 14
<212> PRT
<213> Microcystis aeruginosa
<400> 775
Asp Glu Gln Leu Leu Thr Gln Arg Asn Lys Ile Ala His Gly
1 5 10
<210> 776
<211> 13
<212> PRT
<213> Microcystis aeruginosa
<400> 776
Thr Tyr Asn Leu Val Ile Lys Leu Ile Arg Asp Phe Lys
1 5 10
<210> 777
<211> 11
<212> PRT
<213> Arabidopsis thaliana
<400> 777
Pro Trp Leu Ser Trp Glu Glu Trp Asp Ser Val
1 5 10
<210> 778
<211> 15
<212> PRT
<213> Arabidopsis thaliana
<400> 778
Gly Ser Leu Pro Ala Pro Val Asp Val Thr Cys Ser Leu Ile Glu
1 5 10 15
<210> 779
<211> 9
<212> PRT
<213> Arabidopsis thaliana
<400> 779
Ile Ala Asp Ala Ala Arg Ala Ile Gly
1 5
<210> 780
<211> 15
<212> PRT
<213> Arabidopsis thaliana
<400> 780
Ile Pro Arg Lys Leu Ile Asp Leu Arg His Glu Gly Ser His Arg
1 5 10 15
<210> 781
<211> 13
<212> PRT
<213> Arabidopsis thaliana
<400> 781
Ala Ala Asp Glu Ala Leu Glu Trp Leu Lys Ser Tyr Tyr
1 5 10
<210> 782
<211> 11
<212> PRT
<213> Homo sapiens
<400> 782
Ala Trp Leu Ser Arg Ala Glu Trp Asp Gln Val
1 5 10
<210> 783
<211> 16
<212> PRT
<213> Homo sapiens
<400> 783
Gly Asn Glu Leu Pro Leu Ala Val Ala Ser Thr Ala Asp Leu Ile Arg
1 5 10 15
<210> 784
<211> 9
<212> PRT
<213> Homo sapiens
<400> 784
Leu Lys Cys Leu Ala Gln Glu Val Asn
1 5
<210> 785
<211> 15
<212> PRT
<213> Homo sapiens
<400> 785
Ile Pro Asp Trp Ile Val Asp Leu Arg His Glu Leu Thr His Lys
1 5 10 15
<210> 786
<211> 13
<212> PRT
<213> Homo sapiens
<400> 786
Gly Cys Tyr Phe Val Leu Asp Trp Leu Gln Lys Thr Tyr
1 5 10
<210> 787
<211> 11
<212> PRT
<213> Saccharomyces cerevisiae
<400> 787
Pro Trp Arg Asp Phe Ala Glu Leu Glu Glu Leu
1 5 10
<210> 788
<211> 16
<212> PRT
<213> Saccharomyces cerevisiae
<400> 788
Ser Gln Tyr Leu Pro His Val Val Asp Ser Thr Ala Gln Ile Thr Cys
1 5 10 15
<210> 789
<211> 9
<212> PRT
<213> Saccharomyces cerevisiae
<400> 789
Leu His Thr Leu Ala Ala Lys Ile Gly
1 5
<210> 790
<211> 15
<212> PRT
<213> Saccharomyces cerevisiae
<400> 790
Leu Pro Ser Trp Phe Val Asp Leu Arg His Trp Gly Thr His Glu
1 5 10 15
<210> 791
<211> 13
<212> PRT
<213> Saccharomyces cerevisiae
<400> 791
Ala Ala Asn Glu Ala Leu Ser Trp Leu Tyr Asp His Tyr
1 5 10
<210> 792
<211> 11
<212> PRT
<213> Streptococcus pneumoniae
<400> 792
Ser Lys Pro Cys Ile Glu Ala Glu Asn Met Ile
1 5 10
<210> 793
<211> 16
<212> PRT
<213> Streptococcus pneumoniae
<400> 793
Ala Phe Met Ala Arg Arg Ala Leu Glu Gln Ala Val His Trp Ile Tyr
1 5 10 15
<210> 794
<211> 8
<212> PRT
<213> Streptococcus pneumoniae
<400> 794
Ser Ser Leu Val Trp Asp Asp Asp
1 5
<210> 795
<211> 15
<212> PRT
<213> Streptococcus pneumoniae
<400> 795
Gln Ile Val Leu Leu Ile Arg Trp Gly Asn His Ala Ala His Gly
1 5 10 15
<210> 796
<211> 13
<212> PRT
<213> Streptococcus pneumoniae
<400> 796
Ala Leu His His Leu Tyr Gln Phe Val Asn Phe Ile Asp
1 5 10
<210> 797
<211> 11
<212> PRT
<213> Microcystis aeruginosa
<400> 797
Tyr Asp His Ala Ser Gln Ala Glu Gly Leu Val
1 5 10
<210> 798
<211> 16
<212> PRT
<213> Microcystis aeruginosa
<400> 798
Cys Phe Tyr Thr Arg Phe Val Leu Glu Gln Met Val Cys Trp Leu Tyr
1 5 10 15
<210> 799
<211> 8
<212> PRT
<213> Microcystis aeruginosa
<400> 799
Gly Ala Leu Ile His Glu Gln Thr
1 5
<210> 800
<211> 15
<212> PRT
<213> Microcystis aeruginosa
<400> 800
Lys Ile Arg Thr Ile His Lys Val Gly Asn Asn Ala Ala His Asp
1 5 10 15
<210> 801
<211> 13
<212> PRT
<213> Microcystis aeruginosa
<400> 801
Leu Ile Glu Glu Leu Phe His Leu Thr Tyr Trp Leu Val
1 5 10
<210> 802
<211> 11
<212> PRT
<213> Escherichia coli
<400> 802
Tyr Ala Ile Ala Cys Ala Ala Glu Asn Asn Tyr
1 5 10
<210> 803
<211> 16
<212> PRT
<213> Escherichia coli
<400> 803
Leu Ile Lys Met Arg Met Phe Gly Glu Ala Thr Ala Lys His Leu Gly
1 5 10 15
<210> 804
<211> 8
<212> PRT
<213> Escherichia coli
<400> 804
His Asp Leu Leu Arg Glu Leu Gly
1 5
<210> 805
<211> 15
<212> PRT
<213> Escherichia coli
<400> 805
Val Phe His Lys Leu Arg Arg Ile Gly Asn Gln Ala Val His Glu
1 5 10 15
<210> 806
<211> 13
<212> PRT
<213> Escherichia coli
<400> 806
Cys Leu Arg Leu Gly Phe Arg Leu Ala Val Trp Tyr Tyr
1 5 10
<210> 807
<211> 11
<212> PRT
<213> Bradyrhizobium japonicum
<400> 807
Val Gln Lys Leu Ile Lys Ala Ser Gln Leu Ala
1 5 10
<210> 808
<211> 16
<212> PRT
<213> Bradyrhizobium japonicum
<400> 808
Leu Thr Glu Val Arg Arg Ala Met Lys Ala Ala Ala Asp Leu Phe Trp
1 5 10 15
<210> 809
<211> 10
<212> PRT
<213> Bradyrhizobium japonicum
<400> 809
Leu Asn Arg Leu Gln Glu Phe Ala Arg Val
1 5 10
<210> 810
<211> 13
<212> PRT
<213> Bradyrhizobium japonicum
<400> 810
Arg Arg Leu Asn Asp Leu Ala Ser Lys Gly Val His Ala
1 5 10
<210> 811
<211> 13
<212> PRT
<213> Bradyrhizobium japonicum
<400> 811
Ala Glu Ala Arg Gln Gly Leu Val Gly Leu Tyr Phe Phe
1 5 10
<210> 812
<211> 11
<212> PRT
<213> Leptospira meyeri
<400> 812
Leu Pro Lys Phe Ser Ala Ile Tyr Ser Asn Leu
1 5 10
<210> 813
<211> 16
<212> PRT
<213> Leptospira meyeri
<400> 813
Val His Ser Cys Arg Arg Leu Leu Gln Ser Val Ala Asp Lys Leu Met
1 5 10 15
<210> 814
<211> 10
<212> PRT
<213> Leptospira meyeri
<400> 814
Ile Asn Arg Leu Ile Tyr Tyr Ile Glu Thr
1 5 10
<210> 815
<211> 13
<212> PRT
<213> Leptospira meyeri
<400> 815
Asp Ser Val Phe Gln Ala Ser Gln Lys Gly Ser His Ser
1 5 10
<210> 816
<211> 13
<212> PRT
<213> Leptospira meyeri
<400> 816
Gln Glu Ala Asp Arg Tyr Val Ile His Thr Phe Leu Leu
1 5 10
<210> 817
<211> 11
<212> PRT
<213> Bacteroides coprosuis
<400> 817
Val Val Asp Asp Arg Asp Phe Ser Leu Leu Ala
1 5 10
<210> 818
<211> 16
<212> PRT
<213> Bacteroides coprosuis
<400> 818
Leu Asp Arg Leu His Thr Tyr Val Ile Lys Phe Ile Arg Gln Leu Cys
1 5 10 15
<210> 819
<211> 9
<212> PRT
<213> Bacteroides coprosuis
<400> 819
Phe Gly Lys Tyr Val Lys Phe Ile Val
1 5
<210> 820
<211> 16
<212> PRT
<213> Bacteroides coprosuis
<400> 820
Ile Glu Ala Phe Asn Asp Ile Arg Asn Asn Lys Ser Phe Ala His Asp
1 5 10 15
<210> 821
<211> 13
<212> PRT
<213> Bacteroides coprosuis
<400> 821
Tyr Ala Glu Ser Val Leu Ile Phe Asn Asn Val Thr Asn
1 5 10
<210> 822
<211> 10
<212> PRT
<213> Escherichia coli
<400> 822
Asn Val Asn Glu Asn Ile Tyr Gln Ala Leu
1 5 10
<210> 823
<211> 16
<212> PRT
<213> Escherichia coli
<400> 823
Tyr Asp Arg Val His Thr Ala Leu His Ala Ser Leu Arg Gln Met Cys
1 5 10 15
<210> 824
<211> 9
<212> PRT
<213> Escherichia coli
<400> 824
Leu Ser Leu Ile Thr Ala His Leu Lys
1 5
<210> 825
<211> 16
<212> PRT
<213> Escherichia coli
<400> 825
Leu His Gly Ile Asn Asn Leu Arg Asn Asn Tyr Ser Met Ala His Pro
1 5 10 15
<210> 826
<211> 13
<212> PRT
<213> Escherichia coli
<400> 826
Glu Ala Asp Ala Arg Phe Ala Ile Asn Leu Val Arg Ser
1 5 10
<210> 827
<211> 11
<212> PRT
<213> Lactococcus lactis
<400> 827
Ile Met Asn Ile Gly Tyr Val Glu Lys Ile Leu
1 5 10
<210> 828
<211> 16
<212> PRT
<213> Lactococcus lactis
<400> 828
Val Thr Lys Ser Arg Thr Ile Ile Glu Thr Val Phe Ile Ala Ile Leu
1 5 10 15
<210> 829
<211> 9
<212> PRT
<213> Lactococcus lactis
<400> 829
Arg Ser Leu Val Asn Lys Thr Leu Gly
1 5
<210> 830
<211> 16
<212> PRT
<213> Lactococcus lactis
<400> 830
Val Asp Ser Ile Thr Thr Met Arg Asn Ile Asn Ser Asp Ser His Gly
1 5 10 15
<210> 831
<211> 13
<212> PRT
<213> Lactococcus lactis
<400> 831
Glu Ala Glu Ala Glu Leu Ile Leu Asn Ser Ala Val Asn
1 5 10
<210> 832
<211> 11
<212> PRT
<213> Peptoniphilus indolicus
<400> 832
Phe Leu Tyr Leu Lys Thr Leu Lys Asn Lys Glu
1 5 10
<210> 833
<211> 16
<212> PRT
<213> Peptoniphilus indolicus
<400> 833
Arg Gly Ile Thr Pro Leu Val Thr Glu Leu Phe Ile Leu Ile Ile Asp
1 5 10 15
<210> 834
<211> 9
<212> PRT
<213> Peptoniphilus indolicus
<400> 834
Leu Ile Glu Ile Ile Lys Asn Glu Arg
1 5
<210> 835
<211> 15
<212> PRT
<213> Peptoniphilus indolicus
<400> 835
Ile Arg Asp Val Glu Gly Lys Leu Arg Asn Arg Ala Ala His Glu
1 5 10 15
<210> 836
<211> 13
<212> PRT
<213> Peptoniphilus indolicus
<400> 836
Gly Asn Asn His Tyr Asp Ser Tyr Asp Leu Met Asn Lys
1 5 10
<210> 837
<211> 11
<212> PRT
<213> Mycobacterium tuberculosis
<400> 837
Ile Ser Ala Leu Ala Leu Leu Ala Lys Arg Glu
1 5 10
<210> 838
<211> 16
<212> PRT
<213> Mycobacterium tuberculosis
<400> 838
Arg Ser Ala Thr Pro Ala Ile Thr Ile Val Leu Arg Ala Ala Val Ala
1 5 10 15
<210> 839
<211> 9
<212> PRT
<213> Mycobacterium tuberculosis
<400> 839
Trp Leu Ala Leu Leu Arg Gln Phe Ala
1 5
<210> 840
<211> 15
<212> PRT
<213> Mycobacterium tuberculosis
<400> 840
Leu Gly Arg Phe Glu Ser Arg Val Arg Asn Thr Ala Ala His Glu
1 5 10 15
<210> 841
<211> 11
<212> PRT
<213> Mycobacterium tuberculosis
<400> 841
Ala Asp Leu Thr Leu Tyr Asp Arg Leu Asn Asp
1 5 10
<210> 842
<211> 12
<212> PRT
<213> Streptococcus thermophilus
<400> 842
Tyr Leu Met Ile Asp Val Leu Lys Glu Arg Glu His
1 5 10
<210> 843
<211> 16
<212> PRT
<213> Streptococcus thermophilus
<400> 843
Ile Glu Glu Ile Ile Lys Lys Asp His Glu Gly Leu Ile Val Phe Asp
1 5 10 15
<210> 844
<211> 9
<212> PRT
<213> Streptococcus thermophilus
<400> 844
Tyr Leu Asn Ile Leu Glu Phe Tyr Glu
1 5
<210> 845
<211> 14
<212> PRT
<213> Streptococcus thermophilus
<400> 845
Ile Leu Ser Leu Asn Gly Glu Arg Asn Lys Val Ala His Gly
1 5 10
<210> 846
<211> 13
<212> PRT
<213> Streptococcus thermophilus
<400> 846
Asp Ser Ser Tyr Phe Asn Tyr Tyr Asp Lys Gln Asn Lys
1 5 10
<210> 847
<211> 10
<212> PRT
<213> Synechocystis sp.
<400> 847
Leu Ile Ser Val Val Ala Phe Arg Leu Gly
1 5 10
<210> 848
<211> 16
<212> PRT
<213> Synechocystis sp.
<400> 848
Ile Leu Asp His Arg Lys Gln Ile Asn Phe Ala Leu Asn Asn Gly Gly
1 5 10 15
<210> 849
<211> 10
<212> PRT
<213> Synechocystis sp.
<400> 849
Thr Glu Ile Arg Asn Asp Leu Ala His Cys
1 5 10
<210> 850
<211> 12
<212> PRT
<213> Synechocystis sp.
<400> 850
Asn Lys Ile Phe Pro Gln Leu Glu Glu Ile Ala Asn
1 5 10
<210> 851
<211> 5
<212> PRT
<213> Methanocaldococcus jannaschii
<400> 851
Lys Asn Thr Leu Phe
1 5
<210> 852
<211> 14
<212> PRT
<213> Methanocaldococcus jannaschii
<400> 852
Lys Glu Asn Pro Asn Ser Gln Tyr Ile Lys Asn Glu Ile Ser
1 5 10
<210> 853
<211> 15
<212> PRT
<213> Methanocaldococcus jannaschii
<400> 853
Glu Asn Ile Asp Lys Phe Lys Ile Arg Asn Phe Leu Ala His Ala
1 5 10 15
<210> 854
<211> 13
<212> PRT
<213> Methanocaldococcus jannaschii
<400> 854
Ser Glu Lys Thr Ser Leu Arg Tyr Asn Lys Asn Tyr Ile
1 5 10
<210> 855
<211> 11
<212> PRT
<213> Pyrococcus furiosus
<400> 855
Ser Lys Ile Phe Glu Ser Leu Pro Arg Ile Gly
1 5 10
<210> 856
<211> 15
<212> PRT
<213> Pyrococcus furiosus
<400> 856
Arg Gln Val Glu Trp Leu Arg Asn Leu Val Tyr Gly Arg Leu Trp
1 5 10 15
<210> 857
<211> 15
<212> PRT
<213> Pyrococcus furiosus
<400> 857
Thr Ile Glu Ser Pro Asn Val Val Arg Asn Phe Ile Ala His Ser
1 5 10 15
<210> 858
<211> 13
<212> PRT
<213> Pyrococcus furiosus
<400> 858
Asp Lys Glu Lys Ala Ala Asn Leu Ala Tyr Glu Ala Leu
1 5 10
<210> 859
<211> 5
<212> PRT
<213> Sulfolobus solfataricus
<400> 859
Ala Glu Thr Tyr Ala
1 5
<210> 860
<211> 13
<212> PRT
<213> Sulfolobus solfataricus
<400> 860
Asp Lys Val Thr Arg Ala Ile Ile Glu Asn Glu Val Asp
1 5 10
<210> 861
<211> 13
<212> PRT
<213> Sulfolobus solfataricus
<400> 861
Gly Lys Gly Phe Asp Lys Arg Ile Leu Tyr Ala His Gly
1 5 10
<210> 862
<211> 10
<212> PRT
<213> Sulfolobus solfataricus
<400> 862
Asp Lys Ile Asp Glu Ile Glu Arg Gln Ile
1 5 10
<210> 863
<211> 12
<212> PRT
<213> Desulfococcus oleovorans
<400> 863
Phe Ala Asn Ala Glu Arg Arg Phe Asp Glu Gly Lys
1 5 10
<210> 864
<211> 16
<212> PRT
<213> Desulfococcus oleovorans
<400> 864
Val Leu Arg Leu Tyr Arg Ile Val Glu Met Ala Gly Gln Gln Arg Leu
1 5 10 15
<210> 865
<211> 9
<212> PRT
<213> Desulfococcus oleovorans
<400> 865
Gly Tyr Ser Leu Leu Lys Glu Met Gly
1 5
<210> 866
<211> 17
<212> PRT
<213> Desulfococcus oleovorans
<400> 866
Ser Phe Leu Lys Ile Gln Asp Ser Arg Asn His Ser Phe Leu Ala His
1 5 10 15
Gly
<210> 867
<211> 13
<212> PRT
<213> Desulfococcus oleovorans
<400> 867
Tyr Met Ser Leu Arg Asp Phe Ile Val Ser Leu Asn Ile
1 5 10
<210> 868
<211> 12
<212> PRT
<213> Oscillochloris trichoides
<400> 868
Leu Arg Asn Ala Glu Arg Arg Ala Ala Arg Ala Arg
1 5 10
<210> 869
<211> 16
<212> PRT
<213> Oscillochloris trichoides
<400> 869
Val Ala Arg Leu Tyr Arg Ala Thr Glu Leu Phe Ala Gln Ile Arg Leu
1 5 10 15
<210> 870
<211> 9
<212> PRT
<213> Oscillochloris trichoides
<400> 870
Ser Tyr Ala Leu Leu Gly Lys Leu Asp
1 5
<210> 871
<211> 17
<212> PRT
<213> Oscillochloris trichoides
<400> 871
Pro Leu Asn Asn Ala Leu Thr Arg Arg Asn Gln Ser Ile Leu Ala His
1 5 10 15
Gly
<210> 872
<211> 13
<212> PRT
<213> Oscillochloris trichoides
<400> 872
Tyr His Asp Leu Ala Ser His Leu Tyr Thr Leu Ile Asn
1 5 10
<210> 873
<211> 12
<212> PRT
<213> Homo sapiens
<400> 873
Phe Pro Glu Ile Phe Asp Ala Leu Glu Ser Leu Gln
1 5 10
<210> 874
<211> 14
<212> PRT
<213> Homo sapiens
<400> 874
Lys Leu Thr Ser Cys Leu Glu Arg Ala Leu Gly Asp Val Phe
1 5 10
<210> 875
<211> 9
<212> PRT
<213> Homo sapiens
<400> 875
Ser Glu Glu Leu Ala Gln Val Phe Ser
1 5
<210> 876
<211> 15
<212> PRT
<213> Homo sapiens
<400> 876
Gly Ser Pro Cys Gly Leu Asn Leu Arg Asn Val Leu Trp His Gly
1 5 10 15
<210> 877
<211> 13
<212> PRT
<213> Homo sapiens
<400> 877
Tyr Cys Ser Met Met Ile Leu Leu Thr Ala Gly Leu Gly
1 5 10
<210> 878
<211> 12
<212> PRT
<213> Entamoeba histolytica
<400> 878
Trp Phe Glu Ser Phe Gln Glu Ile Ile Gln Thr Pro
1 5 10
<210> 879
<211> 14
<212> PRT
<213> Entamoeba histolytica
<400> 879
Leu Leu Ser Val Gln Phe Asn Val His Leu Lys Asp Asn Ile
1 5 10
<210> 880
<211> 9
<212> PRT
<213> Entamoeba histolytica
<400> 880
Lys Met Tyr Glu Glu His Thr Val Pro
1 5
<210> 881
<211> 15
<212> PRT
<213> Entamoeba histolytica
<400> 881
Gly Pro Pro Thr Gly Leu Asn Leu Arg Asn Leu Leu Trp His Gly
1 5 10 15
<210> 882
<211> 13
<212> PRT
<213> Entamoeba histolytica
<400> 882
His Ile Cys Leu Leu Ile Ile Leu Tyr Gln Thr Ile Gln
1 5 10
<210> 883
<211> 12
<212> PRT
<213> Staphylococcus aureus
<400> 883
Ile Glu His Gly Ile Ser Arg Phe Leu Glu Lys Asp
1 5 10
<210> 884
<211> 14
<212> PRT
<213> Staphylococcus aureus
<400> 884
Ile Leu Val Pro Gln Phe Glu Ser Thr Val Arg Arg Met Phe
1 5 10
<210> 885
<211> 9
<212> PRT
<213> Staphylococcus aureus
<400> 885
Arg Asp Asp Val Lys Ser Thr Leu Gly
1 5
<210> 886
<211> 15
<212> PRT
<213> Staphylococcus aureus
<400> 886
Val Glu Gln Ser Gly Leu Asn Leu Arg Asn Glu Ile Ala His Gly
1 5 10 15
<210> 887
<211> 12
<212> PRT
<213> Staphylococcus aureus
<400> 887
Lys Cys Ile Leu Val Ile Tyr Leu Phe Leu Ile Leu
1 5 10
<210> 888
<211> 12
<212> PRT
<213> Cyanothece sp.
<400> 888
Leu Leu Lys Gly Ile Gln Ala Tyr Leu Glu Glu Asp
1 5 10
<210> 889
<211> 14
<212> PRT
<213> Cyanothece sp.
<400> 889
Leu Leu Ile Pro Gln Ile Glu Ala Ala Ile Arg Asn Leu Val
1 5 10
<210> 890
<211> 9
<212> PRT
<213> Cyanothece sp.
<400> 890
Ser Glu Gln Val Lys Gln Ser Leu Gly
1 5
<210> 891
<211> 15
<212> PRT
<213> Cyanothece sp.
<400> 891
Thr Asp Gln Arg Gly Trp Asn Val Arg Asn Asn Val Cys His Gly
1 5 10 15
<210> 892
<211> 12
<212> PRT
<213> Cyanothece sp.
<400> 892
Leu Thr Glu Arg Leu Ile His Ile Leu Leu Ile Leu
1 5 10
<210> 893
<211> 9
<212> PRT
<213> Sulfolobus solfataricus
<400> 893
Ile Ser Thr Ser Ala Glu Val Tyr Tyr
1 5
<210> 894
<211> 16
<212> PRT
<213> Sulfolobus solfataricus
<400> 894
Cys Glu Lys Tyr Tyr Lys Ala Ala Glu Glu Ala Ile Lys Leu Leu Val
1 5 10 15
<210> 895
<211> 8
<212> PRT
<213> Sulfolobus solfataricus
<400> 895
Lys Leu Leu Arg Ser Asn Asn Thr
1 5
<210> 896
<211> 15
<212> PRT
<213> Sulfolobus solfataricus
<400> 896
Leu Trp Lys Ser Ala Trp Thr Leu His Val Glu Gly Phe His Glu
1 5 10 15
<210> 897
<211> 13
<212> PRT
<213> Sulfolobus solfataricus
<400> 897
Leu Lys Glu Asp Val Arg Lys Leu Val Ile Phe Ala Val
1 5 10
<210> 898
<211> 9
<212> PRT
<213> Pyrobaculum aerophilum
<400> 898
Tyr Ala Glu Ala Ala Arg Glu Leu Leu
1 5
<210> 899
<211> 16
<212> PRT
<213> Pyrobaculum aerophilum
<400> 899
Ser Glu Lys Ala Trp Gly Ala Ala Ala Leu Ala Val Lys Ala Tyr Ala
1 5 10 15
<210> 900
<211> 7
<212> PRT
<213> Pyrobaculum aerophilum
<400> 900
Lys Ile Ala Gly Glu Leu Gly
1 5
<210> 901
<211> 14
<212> PRT
<213> Pyrobaculum aerophilum
<400> 901
Ala Trp Ala Gln Ala Asn Ala Met His Ile Asn Phe Tyr Glu
1 5 10
<210> 902
<211> 13
<212> PRT
<213> Pyrobaculum aerophilum
<400> 902
Ala Leu Lys Lys Val Ser Arg Leu Val Glu Glu Leu Thr
1 5 10
<210> 903
<211> 11
<212> PRT
<213> Homo sapiens
<400> 903
Arg Arg Trp Leu Arg Gln Ala Arg Ala Asn Phe
1 5 10
<210> 904
<211> 16
<212> PRT
<213> Homo sapiens
<400> 904
Asn Glu Trp Val Cys Phe Lys Cys Tyr Leu Ser Thr Lys Leu Ala Leu
1 5 10 15
<210> 905
<211> 8
<212> PRT
<213> Homo sapiens
<400> 905
Ala Gln Lys Ile Glu Glu Tyr Ser
1 5
<210> 906
<211> 15
<212> PRT
<213> Homo sapiens
<400> 906
Val His Thr Leu Glu Ala Tyr Gly Val Asp Ser Leu Lys Thr Arg
1 5 10 15
<210> 907
<211> 13
<212> PRT
<213> Homo sapiens
<220>
<221> MOD_RES
<222> (2)..(2)
<223> Any amino acid
<400> 907
Val Xaa Glu Cys Thr Ala Cys Ile Ile Ile Lys Leu Glu
1 5 10
<210> 908
<211> 11
<212> PRT
<213> Heamophilus influenzae
<400> 908
Lys Leu Asn Leu Asn Val Leu Asp Ala Ala Phe
1 5 10
<210> 909
<211> 16
<212> PRT
<213> Heamophilus influenzae
<220>
<221> MOD_RES
<222> (14)..(15)
<223> Any amino acid
<400> 909
Ile Gln Lys Phe Glu Phe Val Tyr Glu Leu Ser Leu Lys Xaa Xaa Lys
1 5 10 15
<210> 910
<211> 8
<212> PRT
<213> Heamophilus influenzae
<400> 910
Leu Arg Glu Ala Leu Arg Phe Gly
1 5
<210> 911
<211> 15
<212> PRT
<213> Heamophilus influenzae
<220>
<221> MOD_RES
<222> (8)..(8)
<223> Any amino acid
<400> 911
Lys Trp Val Ala Tyr Arg Asp Xaa Arg Asn Ile Thr Ser His Thr
1 5 10 15
<210> 912
<211> 13
<212> PRT
<213> Heamophilus influenzae
<400> 912
Asp Phe Leu Ile Glu Ser Ser Phe Leu Leu Glu Gln Leu
1 5 10
<210> 913
<211> 6
<212> PRT
<213> Thermus thermophilus
<220>
<221> MOD_RES
<222> (1)..(1)
<223> Any amino acid
<400> 913
Xaa Ala Glu Lys Ala Leu
1 5
<210> 914
<211> 16
<212> PRT
<213> Thermus thermophilus
<400> 914
Ile Gln Arg Phe Glu Tyr Thr Phe Glu Ala Phe Trp Lys Ala Leu Gln
1 5 10 15
<210> 915
<211> 8
<212> PRT
<213> Thermus thermophilus
<400> 915
Ile Arg Leu Ala Arg Glu Val Gly
1 5
<210> 916
<211> 15
<212> PRT
<213> Thermus thermophilus
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<400> 916
Leu Ala Leu Gly Xaa Val Asp Asp Arg Ser Leu Thr Val His Thr
1 5 10 15
<210> 917
<211> 13
<212> PRT
<213> Thermus thermophilus
<220>
<221> MOD_RES
<222> (12)..(12)
<223> Any amino acid
<400> 917
Ile Phe Arg Arg Leu Pro Asp Tyr Ala Arg Leu Xaa Glu
1 5 10
<210> 918
<211> 11
<212> PRT
<213> Rhodococcus equi
<400> 918
Val Asn Leu Leu Arg Arg Ala Asp Gly Leu Leu
1 5 10
<210> 919
<211> 16
<212> PRT
<213> Rhodococcus equi
<400> 919
Phe Cys Ala Ala Tyr Val Gly Ala Leu Arg Gly Ala Ala Ala Val Leu
1 5 10 15
<210> 920
<211> 8
<212> PRT
<213> Rhodococcus equi
<400> 920
Trp Val Leu Met Ala Arg Ala Glu
1 5
<210> 921
<211> 15
<212> PRT
<213> Rhodococcus equi
<400> 921
Tyr Phe Ala Gly Tyr Ser Gly Leu Arg Ala Asp Leu Glu Ala Gly
1 5 10 15
<210> 922
<211> 13
<212> PRT
<213> Rhodococcus equi
<400> 922
Asp Ala Glu Glu Val Asp Gly Phe Tyr Ala Glu Val Gly
1 5 10
<210> 923
<211> 11
<212> PRT
<213> Streptomyces avermitilis
<400> 923
Leu Asp Leu Leu Ala Gln Ala Arg Ala Gly Leu
1 5 10
<210> 924
<211> 16
<212> PRT
<213> Streptomyces avermitilis
<400> 924
Tyr Ala Thr Ala His Leu Ala Ala Leu Arg Thr Ala Ala Ala Val Leu
1 5 10 15
<210> 925
<211> 8
<212> PRT
<213> Streptomyces avermitilis
<400> 925
Trp Glu Val Leu Pro Glu Ile Ala
1 5
<210> 926
<211> 15
<212> PRT
<213> Streptomyces avermitilis
<400> 926
Leu Phe Ala Ser Gly Ala Gly Arg Arg Ala Arg Ala Glu Ala Gly
1 5 10 15
<210> 927
<211> 13
<212> PRT
<213> Streptomyces avermitilis
<400> 927
Ser Asn Arg Asp Ala Asp Asp Leu Ile Arg Asp Val Ala
1 5 10
<210> 928
<211> 11
<212> PRT
<213> Staphylococcus aureus
<400> 928
Ala Leu Ile Val Glu Glu Leu Phe Glu Tyr Ala
1 5 10
<210> 929
<211> 16
<212> PRT
<213> Staphylococcus aureus
<400> 929
Pro Ser Leu Thr Val Gln Val Ala Met Ala Gly Ala Met Leu Ile Gly
1 5 10 15
<210> 930
<211> 8
<212> PRT
<213> Staphylococcus aureus
<400> 930
Thr Glu Ala Val Lys Gln Ser Asp
1 5
<210> 931
<211> 10
<212> PRT
<213> Staphylococcus aureus
<400> 931
His Leu Cys Gln Phe Val Met Ser Gly Gln
1 5 10
<210> 932
<211> 13
<212> PRT
<213> Staphylococcus aureus
<400> 932
Ser Glu Lys Leu Leu Glu Ser Leu Glu Asn Phe Trp Asn
1 5 10
<210> 933
<211> 11
<212> PRT
<213> Enterococcus faecium
<400> 933
Asn Phe Leu Leu Cys Asn Phe Ser Asn Leu Trp
1 5 10
<210> 934
<211> 16
<212> PRT
<213> Enterococcus faecium
<400> 934
Leu Glu Leu Leu Ser Gln Leu Gln Lys Asn Thr Leu Gln Leu Ile Arg
1 5 10 15
<210> 935
<211> 10
<212> PRT
<213> Enterococcus faecium
<400> 935
Lys Lys Phe Ala Lys Thr Thr Ala Arg Leu
1 5 10
<210> 936
<211> 13
<212> PRT
<213> Enterococcus faecium
<400> 936
Lys Val Glu Leu Phe Glu Ala Tyr Lys Asn Ser Leu Leu
1 5 10
<210> 937
<211> 9
<212> PRT
<213> Escherichia coli
<400> 937
Gly Val Tyr Ala Asn Glu Leu Arg Ala
1 5
<210> 938
<211> 16
<212> PRT
<213> Escherichia coli
<400> 938
Gly Gly Ile Arg Glu Ile Glu Phe Ile Val Gln Val Phe Gln Leu Ile
1 5 10 15
<210> 939
<211> 8
<212> PRT
<213> Escherichia coli
<400> 939
Thr Leu Ser Ala Ile Ala Glu Leu
1 5
<210> 940
<211> 15
<212> PRT
<213> Escherichia coli
<400> 940
Glu Gln Leu Arg Val Ala Tyr Leu Phe Leu Arg Arg Leu Glu Asn
1 5 10 15
<210> 941
<211> 13
<212> PRT
<213> Escherichia coli
<400> 941
Leu Thr Gly His Met Thr Asn Val Arg Arg Val Phe Asn
1 5 10
<210> 942
<211> 11
<212> PRT
<213> Sebaldella termitidis
<400> 942
Ser Arg Cys Met Lys Ile Ala Gln Ser Gly Gln
1 5 10
<210> 943
<211> 16
<212> PRT
<213> Sebaldella termitidis
<400> 943
Ile Ala Glu Ala Glu Phe Ile Asn Glu Ser Ile Tyr Met Ile Tyr Leu
1 5 10 15
<210> 944
<211> 8
<212> PRT
<213> Sebaldella termitidis
<400> 944
Lys Asp Met Gln Phe Leu Pro Ile
1 5
<210> 945
<211> 14
<212> PRT
<213> Sebaldella termitidis
<400> 945
Asn Leu Leu Asn Asn Leu Ile Ser Ile Gln Asn Ser Glu Lys
1 5 10
<210> 946
<211> 13
<212> PRT
<213> Sebaldella termitidis
<400> 946
Ala Glu Lys Ile Cys Gly Leu Ile Ile Asn Glu Leu Lys
1 5 10
<210> 947
<211> 10
<212> PRT
<213> Streptomyces coelicolor
<400> 947
Ala Arg Leu Asp Ala Tyr Ala Asn Ser His
1 5 10
<210> 948
<211> 16
<212> PRT
<213> Streptomyces coelicolor
<400> 948
Leu Asp Ala Ala Asp Ser Ile Gly Phe Leu Leu Glu Leu Leu Phe Ala
1 5 10 15
<210> 949
<211> 8
<212> PRT
<213> Streptomyces coelicolor
<400> 949
Trp Glu Leu Asp Arg Phe Pro Leu
1 5
<210> 950
<211> 14
<212> PRT
<213> Streptomyces coelicolor
<400> 950
Glu Leu Leu Ala Thr Leu Gly Arg Ile Thr Gly Ala Gly Gly
1 5 10
<210> 951
<211> 13
<212> PRT
<213> Streptomyces coelicolor
<400> 951
Gln Arg Glu Leu Phe Gly Arg Val Glu Ala Ala Ala Arg
1 5 10
<210> 952
<211> 11
<212> PRT
<213> Flavobacterium psychrophilum
<400> 952
Tyr Ser Ile Tyr Lys Asn Ala Arg Gln Leu Arg
1 5 10
<210> 953
<211> 16
<212> PRT
<213> Flavobacterium psychrophilum
<400> 953
Thr Ser Leu Leu Ile Leu Ser Ser Glu Glu Val Ile Lys Ser Ile Leu
1 5 10 15
<210> 954
<211> 8
<212> PRT
<213> Flavobacterium psychrophilum
<400> 954
Gln Leu Ile Glu Leu Ser Ile Gly
1 5
<210> 955
<211> 15
<212> PRT
<213> Flavobacterium psychrophilum
<400> 955
Lys Leu Thr Glu Phe Asp Asp Lys Lys Asn Gln Gly Phe Tyr Val
1 5 10 15
<210> 956
<211> 13
<212> PRT
<213> Flavobacterium psychrophilum
<400> 956
Lys Thr Glu Phe Thr Glu Thr Lys Val Val Val Asp Arg
1 5 10
<210> 957
<211> 10
<212> PRT
<213> Lactococcus lactis
<400> 957
Lys Cys Ile Asp His Ile Ser Val Leu Ile
1 5 10
<210> 958
<211> 16
<212> PRT
<213> Lactococcus lactis
<400> 958
Thr Phe Ile Ser Ile Thr Ile Ile Glu Glu Val Gly Lys Thr His Ile
1 5 10 15
<210> 959
<211> 8
<212> PRT
<213> Lactococcus lactis
<400> 959
Ser Leu Pro Thr Ile Lys Met Gly
1 5
<210> 960
<211> 14
<212> PRT
<213> Lactococcus lactis
<400> 960
Thr Gly Glu Leu Ile Ser Ile Arg Glu Ser Ser Leu Tyr Ala
1 5 10
<210> 961
<211> 13
<212> PRT
<213> Lactococcus lactis
<400> 961
Lys Glu Gln Ser Arg Ala Leu Leu Leu Tyr Ala Ile Glu
1 5 10
<210> 962
<211> 11
<212> PRT
<213> Pseudomonas putida
<400> 962
Asp Ala Leu Leu Thr Asn Ala Ala Ser Leu Ile
1 5 10
<210> 963
<211> 16
<212> PRT
<213> Pseudomonas putida
<400> 963
Phe Ala Leu Ala His Leu Ala Arg Glu Glu Ile Ala Lys Thr Leu Met
1 5 10 15
<210> 964
<211> 8
<212> PRT
<213> Pseudomonas putida
<400> 964
Thr Ile Asn Ser Ile Val Phe Cys
1 5
<210> 965
<211> 12
<212> PRT
<213> Pseudomonas putida
<400> 965
Phe Arg Asn Asp Leu Lys Asn Asn Ser Leu Tyr Val
1 5 10
<210> 966
<211> 13
<212> PRT
<213> Pseudomonas putida
<400> 966
Ala Glu Arg Ala Leu Arg Thr Ile Thr Leu Ala Trp Asp
1 5 10
<210> 967
<211> 11
<212> PRT
<213> Selenomonas sputigena
<400> 967
Gln Ile Ala Tyr Tyr Leu Tyr Phe Met Tyr Leu
1 5 10
<210> 968
<211> 16
<212> PRT
<213> Selenomonas sputigena
<400> 968
Met Thr Ser Phe Ala Tyr Tyr Lys Ser Tyr Phe Asp Arg Val Thr Ala
1 5 10 15
<210> 969
<211> 10
<212> PRT
<213> Selenomonas sputigena
<400> 969
Arg Leu Cys Glu Phe Tyr Glu Glu Phe Asp
1 5 10
<210> 970
<211> 17
<212> PRT
<213> Selenomonas sputigena
<400> 970
Ile Ile Asp Lys Ala Gln Ala Leu Arg Tyr Ala Asn Pro Leu Thr His
1 5 10 15
Ser
<210> 971
<211> 13
<212> PRT
<213> Selenomonas sputigena
<400> 971
Ile Arg Glu Leu Ser Thr Leu Leu Asp Arg Tyr Ile Ala
1 5 10
<210> 972
<211> 11
<212> PRT
<213> Lactobacillus helveticus
<400> 972
Trp Ile Ser Tyr Tyr Leu Tyr Phe Glu Ser Ile
1 5 10
<210> 973
<211> 16
<212> PRT
<213> Lactobacillus helveticus
<400> 973
Leu Thr Ser Tyr Ala Phe Phe Lys Asn Tyr Phe Asp Arg Thr Thr Ala
1 5 10 15
<210> 974
<211> 10
<212> PRT
<213> Lactobacillus helveticus
<400> 974
Gln Leu Gln Lys Val Tyr Arg Ile Leu Asn
1 5 10
<210> 975
<211> 17
<212> PRT
<213> Lactobacillus helveticus
<400> 975
Ile Ile Ser Lys Ala Asn Asp Leu Arg Asn Asn Asn Pro Leu Ser His
1 5 10 15
Ala
<210> 976
<211> 13
<212> PRT
<213> Lactobacillus helveticus
<400> 976
Ile Ala Thr Met Arg Ser Leu Phe Lys Leu Leu Val Glu
1 5 10
<210> 977
<211> 11
<212> PRT
<213> Lactococcus lactis
<400> 977
Lys Ile Leu Asn Phe Ile Tyr Phe Arg Ala Lys
1 5 10
<210> 978
<211> 16
<212> PRT
<213> Lactococcus lactis
<400> 978
Leu Glu Ser Phe Ala Tyr Tyr Lys Asn Tyr Phe Asp Arg Phe Val Ala
1 5 10 15
<210> 979
<211> 10
<212> PRT
<213> Lactococcus lactis
<400> 979
Lys Leu Ile Asp Gly Leu Lys Gln Leu Asn
1 5 10
<210> 980
<211> 17
<212> PRT
<213> Lactococcus lactis
<400> 980
Ile Ile Asn Glu Ala His Lys Ile Arg Asn Ser Asn Pro Val Ser His
1 5 10 15
Ser
<210> 981
<211> 13
<212> PRT
<213> Lactococcus lactis
<400> 981
Leu Asn Asp Leu Lys Ile Ile Ile Glu Gln Leu Ser Thr
1 5 10
<210> 982
<211> 11
<212> PRT
<213> Pseudomonas sp.
<400> 982
Lys Trp Leu Phe Ile Asp Gln Met Val Asp Leu
1 5 10
<210> 983
<211> 12
<212> PRT
<213> Pseudomonas sp.
<400> 983
Phe Lys Phe Arg Glu Ile Arg Ile Glu Tyr Ser Gln
1 5 10
<210> 984
<211> 9
<212> PRT
<213> Pseudomonas sp.
<400> 984
Tyr Glu Tyr Ala Gln Glu Ile Arg Ser
1 5
<210> 985
<211> 14
<212> PRT
<213> Pseudomonas sp.
<400> 985
Arg Lys Ile Pro Asp Phe Arg Gly Lys Tyr Ala Ala His Ile
1 5 10
<210> 986
<211> 13
<212> PRT
<213> Pseudomonas sp.
<400> 986
Lys Ala Leu Glu Phe Tyr Asn Trp Ile His Ser Asn Glu
1 5 10
<210> 987
<211> 11
<212> PRT
<213> Vibrio paracholerae
<400> 987
Glu Glu Ile Leu Ser Gly Leu Ile Gly Asp Leu
1 5 10
<210> 988
<211> 12
<212> PRT
<213> Vibrio paracholerae
<400> 988
Arg Lys Tyr Val Glu Leu Asn Gln Lys Tyr Gly Lys
1 5 10
<210> 989
<211> 10
<212> PRT
<213> Vibrio paracholerae
<400> 989
Gly Val Tyr Asn Asn Glu Ile Asn Lys Asn
1 5 10
<210> 990
<211> 14
<212> PRT
<213> Vibrio paracholerae
<400> 990
Thr Ala Ile Lys Lys Leu Arg Asn His Cys Val Ala His Val
1 5 10
<210> 991
<211> 13
<212> PRT
<213> Vibrio paracholerae
<400> 991
Phe Ala Asp Glu Phe Leu Asp Trp Ile Cys Pro Asp Asn
1 5 10
<210> 992
<211> 11
<212> PRT
<213> Escherichia coli
<400> 992
Thr Met Ala Asp His Met Val Asn Glu Ala Trp
1 5 10
<210> 993
<211> 16
<212> PRT
<213> Escherichia coli
<400> 993
Phe Asn Leu Ile Leu Gln Ser Ile Glu Phe Arg Leu Lys Gly Leu Ile
1 5 10 15
<210> 994
<211> 10
<212> PRT
<213> Escherichia coli
<400> 994
Lys Val Tyr Asn Thr Phe Ala Ser Lys Ser
1 5 10
<210> 995
<211> 14
<212> PRT
<213> Escherichia coli
<400> 995
Trp Phe Asn Ser Met Arg Ile Leu Arg Asn Arg Phe Met His
1 5 10
<210> 996
<211> 13
<212> PRT
<213> Escherichia coli
<400> 996
Asp Ile Met Pro Glu Leu Ile Phe Thr Ser Val Val Arg
1 5 10
<210> 997
<211> 11
<212> PRT
<213> Geobacter sulfurreducens
<400> 997
Leu Asn Tyr Glu Ala Leu Tyr Val Lys Ser Lys
1 5 10
<210> 998
<211> 16
<212> PRT
<213> Geobacter sulfurreducens
<400> 998
Gln Leu Trp Ala Ser Met Ala Leu Glu Leu Leu Ala Lys Ser Ser Leu
1 5 10 15
<210> 999
<211> 9
<212> PRT
<213> Geobacter sulfurreducens
<400> 999
Gln Arg Leu Gly His Ile Ser Lys Leu
1 5
<210> 1000
<211> 15
<212> PRT
<213> Geobacter sulfurreducens
<400> 1000
Phe Cys Glu Gln Leu Ser Leu Arg Arg Asn Ser Glu Ile His Ser
1 5 10 15
<210> 1001
<211> 13
<212> PRT
<213> Geobacter sulfurreducens
<400> 1001
Asp Ala Trp Glu Val Lys Tyr Trp Tyr Ala Ile Glu Val
1 5 10
<210> 1002
<211> 11
<212> PRT
<213> Streptomyces coelicolor
<400> 1002
Asp Val Ser Tyr Thr Pro Val Ser Asn Gly Met
1 5 10
<210> 1003
<211> 16
<212> PRT
<213> Streptomyces coelicolor
<400> 1003
Val Leu His Leu Gln Ala Ala Thr Glu Val Leu Leu Lys Ala Arg Leu
1 5 10 15
<210> 1004
<211> 10
<212> PRT
<213> Streptomyces coelicolor
<400> 1004
Asp Arg Leu Arg Asp Ile Ala Arg Leu Asp
1 5 10
<210> 1005
<211> 15
<212> PRT
<213> Streptomyces coelicolor
<400> 1005
Arg Ile Lys Glu Pro Gly Glu Ser Arg Asn Ala Leu Gln His Tyr
1 5 10 15
<210> 1006
<211> 13
<212> PRT
<213> Streptomyces coelicolor
<400> 1006
Tyr Ala Ile Glu Ser Arg Ala Ala Arg Val Leu Asp Phe
1 5 10
<210> 1007
<211> 10
<212> PRT
<213> Leptospira interrogans
<400> 1007
Cys Thr Arg Leu Tyr Asn Gln Ile Leu Glu
1 5 10
<210> 1008
<211> 16
<212> PRT
<213> Leptospira interrogans
<400> 1008
Tyr Thr Lys Leu Phe Asn Ile Leu Asp Lys Val Ala Ala Ile Val Tyr
1 5 10 15
<210> 1009
<211> 6
<212> PRT
<213> Leptospira interrogans
<400> 1009
Phe Pro Ser Thr Phe Gly
1 5
<210> 1010
<211> 13
<212> PRT
<213> Leptospira interrogans
<400> 1010
His His Leu Arg Val Arg Arg Asn Asn Ile Val His Trp
1 5 10
<210> 1011
<211> 13
<212> PRT
<213> Leptospira interrogans
<400> 1011
Glu Glu Asp Val Gln Arg Leu Phe Leu Ile Ser Lys Ala
1 5 10
<210> 1012
<211> 10
<212> PRT
<213> Shigella boydii
<400> 1012
Met Glu Met Val Leu Asn Arg Leu Lys Ser
1 5 10
<210> 1013
<211> 16
<212> PRT
<213> Shigella boydii
<400> 1013
Phe Arg Leu Cys Phe Gly Ile Leu Asp Lys Ile Ala Val Ala Ile Cys
1 5 10 15
<210> 1014
<211> 8
<212> PRT
<213> Shigella boydii
<400> 1014
Pro Gln Lys Asn Ile Tyr Phe Gln
1 5
<210> 1015
<211> 15
<212> PRT
<213> Shigella boydii
<400> 1015
Glu Leu Ala Phe Tyr Lys Glu Trp Arg Asn Gly Leu Glu His Lys
1 5 10 15
<210> 1016
<211> 13
<212> PRT
<213> Shigella boydii
<400> 1016
Ile His His Phe Glu His Leu Leu Gln Ile Thr Arg Ser
1 5 10
<210> 1017
<211> 10
<212> PRT
<213> Enterococcus faecalis
<400> 1017
Phe Tyr Ser Leu Phe Asn Gln Ile Lys Gln
1 5 10
<210> 1018
<211> 16
<212> PRT
<213> Enterococcus faecalis
<400> 1018
Tyr Arg Ser Val Tyr Ser Ile Phe Asp Lys Ile Ala Tyr Phe Leu Asn
1 5 10 15
<210> 1019
<211> 8
<212> PRT
<213> Enterococcus faecalis
<400> 1019
Pro Lys Asn Leu Ile Thr Phe His
1 5
<210> 1020
<211> 15
<212> PRT
<213> Enterococcus faecalis
<400> 1020
Asn Leu Glu Lys Ile Ala Glu Ile Arg Asn Ala Met Glu His Lys
1 5 10 15
<210> 1021
<211> 13
<212> PRT
<213> Enterococcus faecalis
<400> 1021
Glu Lys Ile Thr Leu Glu Leu Phe Lys Leu Thr Arg Glu
1 5 10
<210> 1022
<211> 10
<212> PRT
<213> Clostridium thermocellum
<400> 1022
Phe Asn Asn Arg Ala Phe Asp Leu Ile Val
1 5 10
<210> 1023
<211> 16
<212> PRT
<213> Clostridium thermocellum
<400> 1023
Tyr Thr Arg Phe Glu Gly Leu Ile Asp Thr Ile Tyr His Ile Ile Asn
1 5 10 15
<210> 1024
<211> 7
<212> PRT
<213> Clostridium thermocellum
<400> 1024
Lys Pro Ser Ser Glu Phe Arg
1 5
<210> 1025
<211> 15
<212> PRT
<213> Clostridium thermocellum
<400> 1025
Val Tyr Lys Lys Ile Asn Lys Phe Arg Asn Asn Ile Val His Asn
1 5 10 15
<210> 1026
<211> 13
<212> PRT
<213> Clostridium thermocellum
<400> 1026
Tyr Thr Thr Ser Thr Glu Phe Leu Asn Asn Ile Lys Asp
1 5 10
<210> 1027
<211> 10
<212> PRT
<213> Bacillus cereus
<400> 1027
Leu Asn Asn Arg Ile Phe Gln Leu Asp Leu
1 5 10
<210> 1028
<211> 16
<212> PRT
<213> Bacillus cereus
<400> 1028
Phe Pro Lys Ala Phe Thr Ala Leu Asp Leu Leu Ala His Leu Leu Phe
1 5 10 15
<210> 1029
<211> 7
<212> PRT
<213> Bacillus cereus
<400> 1029
Lys Thr Glu Lys Lys Ile Lys
1 5
<210> 1030
<211> 15
<212> PRT
<213> Bacillus cereus
<400> 1030
Glu Phe Gln Lys Ala Ser Lys Val Arg Asn Asp Ile Ile His Asn
1 5 10 15
<210> 1031
<211> 13
<212> PRT
<213> Bacillus cereus
<400> 1031
Tyr Thr Pro Ser Lys Glu Ile Leu Asn Ile Ala Arg Gly
1 5 10
<210> 1032
<211> 10
<212> PRT
<213> Pseudomonas syringae
<400> 1032
Glu Tyr Leu Arg Cys Lys Asp Ala Phe Glu
1 5 10
<210> 1033
<211> 16
<212> PRT
<213> Pseudomonas syringae
<400> 1033
Ser Ser Phe Ile His His Leu Tyr Glu Leu Tyr Met Ala Leu Phe Ala
1 5 10 15
<210> 1034
<211> 8
<212> PRT
<213> Pseudomonas syringae
<400> 1034
Ser Ile Asp Arg Gly Ala Val Ser
1 5
<210> 1035
<211> 16
<212> PRT
<213> Pseudomonas syringae
<400> 1035
Phe Gly Pro Ala Phe Arg Ser Met Arg Asn Lys Ile Ala Gly His Val
1 5 10 15
<210> 1036
<211> 13
<212> PRT
<213> Pseudomonas syringae
<400> 1036
Val Lys Leu Thr Glu Phe Phe Gln Lys Tyr His Pro Tyr
1 5 10
<210> 1037
<211> 10
<212> PRT
<213> Burkholderia xenovorans
<400> 1037
Glu Tyr Leu Arg Cys Asp Asp Ala Leu His
1 5 10
<210> 1038
<211> 16
<212> PRT
<213> Burkholderia xenovorans
<400> 1038
Ala Arg Phe Ile His His Leu Tyr Glu Phe Asn Ile Ala Cys Ala Gln
1 5 10 15
<210> 1039
<211> 8
<212> PRT
<213> Burkholderia xenovorans
<400> 1039
Arg Val Arg Arg Gln Ala Tyr Asn
1 5
<210> 1040
<211> 16
<212> PRT
<213> Burkholderia xenovorans
<400> 1040
Phe Ala Lys Ala Phe Arg Thr Ala Arg Asn Thr Thr Asn Gly His Ala
1 5 10 15
<210> 1041
<211> 13
<212> PRT
<213> Burkholderia xenovorans
<400> 1041
Leu Asn Leu Ser Asp Phe Phe Thr Arg Tyr His Arg Phe
1 5 10
<210> 1042
<211> 10
<212> PRT
<213> Microcystis aeruginosa
<400> 1042
Glu His Leu Asp Cys Glu Leu Trp Glu Arg
1 5 10
<210> 1043
<211> 16
<212> PRT
<213> Microcystis aeruginosa
<400> 1043
Ile Arg Asn Ala Thr Val Ile Leu Glu Asp Arg Met Arg Lys Leu Gly
1 5 10 15
<210> 1044
<211> 8
<212> PRT
<213> Microcystis aeruginosa
<400> 1044
Gly Ile Val Asn Leu Ile Phe Gly
1 5
<210> 1045
<211> 15
<212> PRT
<213> Microcystis aeruginosa
<400> 1045
Tyr Ser Gly Thr Met Lys Ile Phe Arg Asn Arg Tyr Ala His Arg
1 5 10 15
<210> 1046
<211> 13
<212> PRT
<213> Microcystis aeruginosa
<400> 1046
Ile Ile Val Phe Ile Asp Leu Leu Leu Lys Met Leu Asp
1 5 10
<210> 1047
<211> 11
<212> PRT
<213> Vibrio parahaemolyticus
<400> 1047
Ser Arg Asn Val His Pro Asp Val Leu Lys Tyr
1 5 10
<210> 1048
<211> 16
<212> PRT
<213> Vibrio parahaemolyticus
<400> 1048
Val Phe Glu Ala Thr Lys Ser Val Ala Asp Lys Ile Arg Asn Lys Thr
1 5 10 15
<210> 1049
<211> 8
<212> PRT
<213> Vibrio parahaemolyticus
<400> 1049
Val Leu Val Asp Glu Ala Phe Ser
1 5
<210> 1050
<211> 15
<212> PRT
<213> Vibrio parahaemolyticus
<400> 1050
Leu Lys Gly Leu Phe Gly Thr Phe Arg Asn Thr Thr Ala His Ala
1 5 10 15
<210> 1051
<211> 13
<212> PRT
<213> Vibrio parahaemolyticus
<400> 1051
Ile Leu Ser Met Val Ser Leu Val His Arg Arg Leu Asp
1 5 10
<210> 1052
<211> 11
<212> PRT
<213> Lactococcus lactis
<400> 1052
Ala Leu Glu Leu His Ser Glu Val Thr Lys Tyr
1 5 10
<210> 1053
<211> 16
<212> PRT
<213> Lactococcus lactis
<400> 1053
Val Phe Glu Ser Cys Lys Gly Leu Phe Asp Arg Ile Arg Leu Ile Ser
1 5 10 15
<210> 1054
<211> 8
<212> PRT
<213> Lactococcus lactis
<400> 1054
Thr Leu Ile Asn Gln Ala Phe Asn
1 5
<210> 1055
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 1055
Ile Lys Thr Cys Leu Tyr Leu Tyr Arg Asn His Gln Ala His Val
1 5 10 15
<210> 1056
<211> 13
<212> PRT
<213> Lactococcus lactis
<400> 1056
Gly Leu Met Ser Ile Ser Leu Ala His Glu Leu Leu Asp
1 5 10
<210> 1057
<211> 11
<212> PRT
<213> Nematostella vectensis
<400> 1057
Ser Thr Thr Leu Thr Thr Phe Leu Asn Leu His
1 5 10
<210> 1058
<211> 16
<212> PRT
<213> Nematostella vectensis
<400> 1058
Glu Asp Tyr Asp Ile Thr Leu Leu Thr Cys Leu Leu Arg Asn Ile Cys
1 5 10 15
<210> 1059
<211> 8
<212> PRT
<213> Nematostella vectensis
<400> 1059
Asp Lys Leu Pro Pro Ala Tyr Asp
1 5
<210> 1060
<211> 15
<212> PRT
<213> Nematostella vectensis
<400> 1060
Val Val Arg Leu Arg His Tyr Arg Asn Asp Leu Tyr Ala His Ile
1 5 10 15
<210> 1061
<211> 13
<212> PRT
<213> Nematostella vectensis
<400> 1061
Trp Ala Asp Ile Ser Ala Ala Leu Leu Ser Leu Gly Gly
1 5 10
<210> 1062
<211> 11
<212> PRT
<213> Branchiostoma floridae
<400> 1062
Pro Pro Ser Leu Pro Ala Gln Leu Lys Lys His
1 5 10
<210> 1063
<211> 16
<212> PRT
<213> Branchiostoma floridae
<400> 1063
Glu Glu Phe Asp Ile Ser Leu Leu Leu Leu Leu Leu Lys Glu Leu Val
1 5 10 15
<210> 1064
<211> 8
<212> PRT
<213> Branchiostoma floridae
<400> 1064
Gly Arg Asp Ala Pro Tyr Ser Asp
1 5
<210> 1065
<211> 13
<212> PRT
<213> Branchiostoma floridae
<400> 1065
Lys Leu Gly Gln Phe Arg Asn Lys Asn Tyr Gly His Ile
1 5 10
<210> 1066
<211> 13
<212> PRT
<213> Branchiostoma floridae
<400> 1066
Trp Asp Glu Leu Thr Glu Ile Leu Val Asp Leu Gly Gly
1 5 10
<210> 1067
<211> 11
<212> PRT
<213> Homo sapiens
<400> 1067
Pro Pro Leu Leu Lys Lys Glu Leu Leu Ile His
1 5 10
<210> 1068
<211> 16
<212> PRT
<213> Homo sapiens
<400> 1068
Lys Gln Phe Asp Leu Cys Leu Leu Leu Ala Leu Ile Lys His Leu Asn
1 5 10 15
<210> 1069
<211> 8
<212> PRT
<213> Homo sapiens
<400> 1069
Asn Met Glu Pro Pro Ser Ser Asp
1 5
<210> 1070
<211> 15
<212> PRT
<213> Homo sapiens
<400> 1070
Ile Leu Arg Leu Cys Lys Tyr Arg Asp Ile Leu Leu Ser Glu Ile
1 5 10 15
<210> 1071
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1071
Trp Lys Lys Val Ser Asp Ile Leu Leu Arg Leu Gly Met
1 5 10
<210> 1072
<211> 11
<212> PRT
<213> Escherichia coli
<400> 1072
Val Thr Ala Glu Lys Leu Leu Val Ser Gly Leu
1 5 10
<210> 1073
<211> 16
<212> PRT
<213> Escherichia coli
<400> 1073
Leu Tyr Pro Glu Leu Arg Thr Ile Glu Gly Val Leu Lys Ser Lys Met
1 5 10 15
<210> 1074
<211> 8
<212> PRT
<213> Escherichia coli
<400> 1074
Tyr Ile Leu Lys Pro Gln Phe Ala
1 5
<210> 1075
<211> 14
<212> PRT
<213> Escherichia coli
<400> 1075
Ala Tyr Thr Phe Phe Asn Val Glu Arg His Ser Leu Phe His
1 5 10
<210> 1076
<211> 13
<212> PRT
<213> Escherichia coli
<400> 1076
Met Ile Ser Asp Met Ala Arg Leu Met Gly Lys Ala Thr
1 5 10
<210> 1077
<211> 11
<212> PRT
<213> Photobacterium profundum
<400> 1077
Asp Thr Tyr Arg Ser Leu Leu Ser Ser Ser Tyr
1 5 10
<210> 1078
<211> 16
<212> PRT
<213> Photobacterium profundum
<400> 1078
Ile Tyr Pro Asp Leu Arg Val Leu Glu Gly Val Ile Lys Glu Ala Met
1 5 10 15
<210> 1079
<211> 8
<212> PRT
<213> Photobacterium profundum
<400> 1079
Thr Glu Leu Lys Thr Glu Tyr Asn
1 5
<210> 1080
<211> 14
<212> PRT
<213> Photobacterium profundum
<400> 1080
Cys Tyr Ala Tyr Phe Lys Ala His Arg His Ser Leu Phe His
1 5 10
<210> 1081
<211> 13
<212> PRT
<213> Photobacterium profundum
<400> 1081
Thr Thr Asp Thr Ile Gly Glu Val Met Gln Met Ser Glu
1 5 10
<210> 1082
<211> 11
<212> PRT
<213> Geobacillus thermoglucosidasius
<400> 1082
Leu Tyr Asp Arg Asp Arg Ile Glu Ala Ser Glu
1 5 10
<210> 1083
<211> 16
<212> PRT
<213> Geobacillus thermoglucosidasius
<400> 1083
Val Ser Gly Thr Leu Arg Ala Phe Glu Gly Phe Phe Lys Lys Leu Leu
1 5 10 15
<210> 1084
<211> 8
<212> PRT
<213> Geobacillus thermoglucosidasius
<400> 1084
Asp Ile Ser Glu Lys Val Phe Asn
1 5
<210> 1085
<211> 14
<212> PRT
<213> Geobacillus thermoglucosidasius
<400> 1085
Met Leu Asn His Met Ser Gln Asp Arg Asn Pro Tyr Ser His
1 5 10
<210> 1086
<211> 13
<212> PRT
<213> Geobacillus thermoglucosidasius
<400> 1086
Pro Leu Arg Thr Leu Asn Gln Ala Ile Ser Leu His Asn
1 5 10
<210> 1087
<211> 12
<212> PRT
<213> Teredinibacter turnerae
<400> 1087
Cys Arg Ser Ile Arg Lys Leu Leu Asn Met Asn Ala
1 5 10
<210> 1088
<211> 16
<212> PRT
<213> Teredinibacter turnerae
<400> 1088
Ser Tyr Pro Leu Ile Tyr Glu Ile Glu Asn Leu Val Arg Lys Leu Ile
1 5 10 15
<210> 1089
<211> 9
<212> PRT
<213> Teredinibacter turnerae
<400> 1089
Ile Gln Leu Ser Asn Phe Leu Phe Asp
1 5
<210> 1090
<211> 15
<212> PRT
<213> Teredinibacter turnerae
<400> 1090
Arg Trp Gly Lys Leu Tyr Lys Leu Arg Cys Lys Ile Ala His Asn
1 5 10 15
<210> 1091
<211> 13
<212> PRT
<213> Teredinibacter turnerae
<400> 1091
Thr Thr Lys Leu Val Glu Glu Val Lys Leu Lys Ile Leu
1 5 10
<210> 1092
<211> 5
<212> PRT
<213> Methanococcus maripaludis
<400> 1092
Phe Arg Leu Met Tyr
1 5
<210> 1093
<211> 16
<212> PRT
<213> Methanococcus maripaludis
<400> 1093
Phe Leu Asp Ser Val Leu Ala Leu Glu Ile Tyr His Thr Leu Lys Phe
1 5 10 15
<210> 1094
<211> 10
<212> PRT
<213> Methanococcus maripaludis
<400> 1094
Phe Ile Asn Lys Met Lys Asp Val Phe Asn
1 5 10
<210> 1095
<211> 15
<212> PRT
<213> Methanococcus maripaludis
<400> 1095
Ile Cys Arg Ile Ile Arg Asp Thr Arg Asn Lys Leu Val His Asp
1 5 10 15
<210> 1096
<211> 13
<212> PRT
<213> Methanococcus maripaludis
<400> 1096
Pro Tyr Phe Leu Ile Glu Leu Leu Lys Asn Ile Phe Lys
1 5 10
<210> 1097
<211> 11
<212> PRT
<213> Novosphingobium pentaromativorans
<400> 1097
Val His Arg Ala Leu Ser Trp Leu Arg Arg Ala
1 5 10
<210> 1098
<211> 14
<212> PRT
<213> Novosphingobium pentaromativorans
<400> 1098
Phe Ile Leu Leu Trp Ile Gly Phe Asn Ala Ala Tyr Ala Gly
1 5 10
<210> 1099
<211> 10
<212> PRT
<213> Novosphingobium pentaromativorans
<400> 1099
Glu Arg Ser Arg Thr Ala Ile Asn Tyr Ala
1 5 10
<210> 1100
<211> 15
<212> PRT
<213> Novosphingobium pentaromativorans
<400> 1100
Leu Phe Asp Arg Leu Tyr Val Leu Arg Asn Gln Leu Val His Gly
1 5 10 15
<210> 1101
<211> 13
<212> PRT
<213> Novosphingobium pentaromativorans
<400> 1101
Arg Asp Gln Val Arg Asp Gly Ala Ser Leu Leu Gly Cys
1 5 10
<210> 1102
<211> 12
<212> PRT
<213> Chlorobium chlorochromatii
<400> 1102
Ile Met Glu Gln Arg Lys Ala Ile Leu Glu Pro Leu
1 5 10
<210> 1103
<211> 16
<212> PRT
<213> Chlorobium chlorochromatii
<400> 1103
Ala Val Ala Tyr Asn His Phe Val Pro Leu Leu Ala Gln Asp Leu Ile
1 5 10 15
<210> 1104
<211> 6
<212> PRT
<213> Chlorobium chlorochromatii
<400> 1104
Lys Ile Ser Asn Lys Lys
1 5
<210> 1105
<211> 15
<212> PRT
<213> Chlorobium chlorochromatii
<400> 1105
Ser Glu Lys Leu Lys Thr Phe Arg Asp Lys Tyr Tyr Ala His Leu
1 5 10 15
<210> 1106
<211> 13
<212> PRT
<213> Chlorobium chlorochromatii
<400> 1106
Phe Leu Gly Ile His Arg Lys Ser Ala Asn Glu Met Trp
1 5 10
<210> 1107
<211> 12
<212> PRT
<213> Lactococcus lactis
<400> 1107
Asp Ala Tyr Asn Lys Leu Ile Leu Leu Lys Gln Tyr
1 5 10
<210> 1108
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 1108
Phe Phe Tyr Asn Asn Leu Leu Asp Ser Leu Val Ile Ala Ile Phe
1 5 10 15
<210> 1109
<211> 6
<212> PRT
<213> Lactococcus lactis
<400> 1109
Asn Tyr Thr Asn Phe Pro
1 5
<210> 1110
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 1110
Leu Glu Tyr Leu Tyr Ala Gln Arg Asn Lys Ile Tyr Val His Asn
1 5 10 15
<210> 1111
<211> 13
<212> PRT
<213> Lactococcus lactis
<400> 1111
Asn Tyr Ala Trp Glu Pro Thr Asn Ile Asn Asp Trp Glu
1 5 10
<210> 1112
<211> 16
<212> PRT
<213> Escherichia coli
<400> 1112
Glu Ser Val Ile Ala His Met Asn Glu Leu Leu Ile Ala Leu Ser Asp
1 5 10 15
<210> 1113
<211> 15
<212> PRT
<213> Escherichia coli
<400> 1113
Arg Tyr Thr Gln Gln Gln Arg Leu Arg Thr Ala Ile Ala His His
1 5 10 15
<210> 1114
<211> 12
<212> PRT
<213> Escherichia coli
<400> 1114
Glu Ala Arg His Glu Gln Leu Thr Lys Gly Gly Thr
1 5 10
<210> 1115
<211> 16
<212> PRT
<213> Cronobacter sakazakii
<400> 1115
Gln His Val Ile Ala Pro Met Asn Glu Leu Leu Ile Ala Leu Ser Asp
1 5 10 15
<210> 1116
<211> 15
<212> PRT
<213> Cronobacter sakazakii
<400> 1116
Arg Tyr Asp Leu Gln Gln Gln Leu Arg Thr Ala Ile Ala His His
1 5 10 15
<210> 1117
<211> 13
<212> PRT
<213> Cronobacter sakazakii
<400> 1117
Ala Ala Glu Arg Leu Ala Glu Leu Thr Arg Gly Gly Thr
1 5 10
<210> 1118
<211> 16
<212> PRT
<213> Homo sapiens
<400> 1118
Glu Ser Arg Tyr Arg Thr Leu Arg Asn Val Gly Asn Glu Ser Asp Ile
1 5 10 15
<210> 1119
<211> 10
<212> PRT
<213> Homo sapiens
<400> 1119
Leu Gln Pro Gly Pro Ser Glu His Ser Lys
1 5 10
<210> 1120
<211> 14
<212> PRT
<213> Homo sapiens
<400> 1120
Val Gly Asp Leu Leu Lys Phe Ile Arg Asn Leu Gly Glu His
1 5 10
<210> 1121
<211> 13
<212> PRT
<213> Homo sapiens
<400> 1121
Ile Gly Asp Pro Ser Leu Tyr Phe Gln Lys Thr Phe Pro
1 5 10
<210> 1122
<211> 16
<212> PRT
<213> Arabidopsis thaliana
<400> 1122
Glu Met Arg Leu Ser Phe Leu Arg Asp Ala Ser Asp Arg Val Glu Leu
1 5 10 15
<210> 1123
<211> 10
<212> PRT
<213> Arabidopsis thaliana
<400> 1123
Met Glu Ser Thr Ala Pro Val Ala Ile Gly
1 5 10
<210> 1124
<211> 15
<212> PRT
<213> Arabidopsis thaliana
<400> 1124
Ile Arg Asp Leu Leu Arg Val Ile Arg Asn Lys Leu Asn His His
1 5 10 15
<210> 1125
<211> 13
<212> PRT
<213> Arabidopsis thaliana
<400> 1125
Pro Glu Gly Phe Asp Glu Tyr Phe Ala Val Arg Phe Pro
1 5 10
<210> 1126
<211> 12
<212> PRT
<213> Helicobacter pylori
<400> 1126
Tyr Glu Leu Leu Trp Gln Glu Val Ile Arg Ala Lys
1 5 10
<210> 1127
<211> 15
<212> PRT
<213> Helicobacter pylori
<400> 1127
Trp Val Ser Leu Gln Asn Val Met Arg Arg Ile Ile Glu Tyr Tyr
1 5 10 15
<210> 1128
<211> 5
<212> PRT
<213> Helicobacter pylori
<400> 1128
Phe Arg Ile Leu Gly
1 5
<210> 1129
<211> 17
<212> PRT
<213> Helicobacter pylori
<400> 1129
Lys Gln Val Phe Ser Ser Phe Ile Ser Trp Phe Asn Asp Gly Ser His
1 5 10 15
Gly
<210> 1130
<211> 13
<212> PRT
<213> Helicobacter pylori
<400> 1130
Ile Glu Thr Tyr Leu Lys Val Phe Glu Asn Ile Phe Lys
1 5 10
<210> 1131
<211> 12
<212> PRT
<213> Streptococcus mutans
<400> 1131
His Leu Met Leu Val Asp Glu Leu Lys Lys Ala Ile
1 5 10
<210> 1132
<211> 15
<212> PRT
<213> Streptococcus mutans
<400> 1132
Glu Lys Tyr His Phe Asn Leu Leu Arg Asn Leu Leu Glu Lys Thr
1 5 10 15
<210> 1133
<211> 5
<212> PRT
<213> Streptococcus mutans
<400> 1133
Ala Thr Phe Leu Gly
1 5
<210> 1134
<211> 14
<212> PRT
<213> Streptococcus mutans
<400> 1134
Pro Ala Pro Tyr Ile Arg Arg Ile Asn Leu His Ser His Ser
1 5 10
<210> 1135
<211> 13
<212> PRT
<213> Streptococcus mutans
<400> 1135
Lys Lys Val Leu Glu Arg Val Phe Asn Gln Phe Leu Gln
1 5 10
<210> 1136
<211> 12
<212> PRT
<213> Escherichia coli
<400> 1136
His Leu His Leu Lys Gln Thr Ile Glu Gln Ala Ile
1 5 10
<210> 1137
<211> 15
<212> PRT
<213> Escherichia coli
<400> 1137
Glu Arg Tyr His Phe Thr Leu Leu Arg Asn Leu Tyr Glu Lys Thr
1 5 10 15
<210> 1138
<211> 5
<212> PRT
<213> Escherichia coli
<400> 1138
Ala Ser Phe Leu Gly
1 5
<210> 1139
<211> 13
<212> PRT
<213> Escherichia coli
<400> 1139
Leu Tyr Leu Ser Arg Ile Ile Asn Phe Thr Ser His Ser
1 5 10
<210> 1140
<211> 13
<212> PRT
<213> Escherichia coli
<400> 1140
Lys Ala Thr Val Lys Leu Leu Leu Asp His Leu Lys Asn
1 5 10
<210> 1141
<211> 12
<212> PRT
<213> Bacteroides fragilis
<400> 1141
Lys Glu Ile Glu Glu Glu Arg Thr Val Gln Asn Ile
1 5 10
<210> 1142
<211> 16
<212> PRT
<213> Bacteroides fragilis
<400> 1142
Thr Ser Phe Gly Glu Val Thr Glu Glu Tyr His Asp Glu Leu Tyr Ser
1 5 10 15
<210> 1143
<211> 8
<212> PRT
<213> Bacteroides fragilis
<400> 1143
Tyr Ile Lys Glu Leu Ser Asn Gly
1 5
<210> 1144
<211> 15
<212> PRT
<213> Bacteroides fragilis
<400> 1144
Gln Lys Thr Leu Thr Glu Lys Ile Arg His Gln Ile His His Pro
1 5 10 15
<210> 1145
<211> 13
<212> PRT
<213> Bacteroides fragilis
<400> 1145
Glu Thr Glu Ile Arg Gln Ser Ile Glu Asp Met Arg Ala
1 5 10
<210> 1146
<211> 12
<212> PRT
<213> Methylobacillus flagellates
<400> 1146
Ser Asn Gln Ile Pro Thr Arg Val Ser Pro Val Leu
1 5 10
<210> 1147
<211> 16
<212> PRT
<213> Methylobacillus flagellates
<400> 1147
Ser Ala Phe Gly Glu Ala Ser Tyr Glu Tyr His Asn Glu Leu Tyr Gly
1 5 10 15
<210> 1148
<211> 8
<212> PRT
<213> Methylobacillus flagellates
<400> 1148
Tyr Asn Arg Leu Arg Arg Asp Gly
1 5
<210> 1149
<211> 15
<212> PRT
<213> Methylobacillus flagellates
<400> 1149
Gln Val Ile Leu Thr Glu Tyr Ile Arg His Gln Ile His His Pro
1 5 10 15
<210> 1150
<211> 13
<212> PRT
<213> Methylobacillus flagellates
<400> 1150
Thr Ala Glu Leu Thr Glu Ser Ile Glu Thr Met Arg Leu
1 5 10
<210> 1151
<211> 12
<212> PRT
<213> Campylobacter hominis
<400> 1151
Lys Asp Gly Glu Gln Lys Lys Glu Val Lys Asn Val
1 5 10
<210> 1152
<211> 16
<212> PRT
<213> Campylobacter hominis
<400> 1152
Met Ala Phe Gly Glu Ile Thr Glu Glu Tyr His Asn Glu Leu Tyr Gly
1 5 10 15
<210> 1153
<211> 8
<212> PRT
<213> Campylobacter hominis
<400> 1153
Tyr Lys Lys Leu Lys Lys Asp Gly
1 5
<210> 1154
<211> 15
<212> PRT
<213> Campylobacter hominis
<400> 1154
Lys Leu Thr Leu Thr Glu Tyr Ile Arg His Gln Ile His His Pro
1 5 10 15
<210> 1155
<211> 13
<212> PRT
<213> Campylobacter hominis
<400> 1155
Leu Ser Glu Leu Lys Asp Ser Ile Glu Met Met Arg Asn
1 5 10
<210> 1156
<211> 15
<212> PRT
<213> Homo sapiens
<400> 1156
Ile Pro Asp Trp Ile Val Asp Leu Arg His Glu Leu Thr His Lys
1 5 10 15
<210> 1157
<211> 15
<212> PRT
<213> Mycobacterium tuberculosis
<400> 1157
Leu Gly Arg Phe Glu Ser Arg Val Arg Asn Thr Ala Ala His Glu
1 5 10 15
<210> 1158
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 1158
Trp Ile Arg Ala Gly Trp Phe Ile Arg Asn Arg Ser Ala His Tyr
1 5 10 15
<210> 1159
<211> 15
<212> PRT
<213> Thermus thermophilus
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<400> 1159
Leu Ala Leu Gly Xaa Val Asp Asp Arg Ser Leu Thr Val His Thr
1 5 10 15
<210> 1160
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1160
Leu Lys Ser Met Leu Tyr Ser Met Arg Asn Ser Ser Phe His Phe Ser
1 5 10 15
Thr Glu Asn Val Asp Asn
20
<210> 1161
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1161
Leu Lys Asp Val Ile Tyr Ser Met Arg Asn Asp Ser Phe His Tyr Ala
1 5 10 15
Thr Glu Asn His Asn Asn
20
<210> 1162
<211> 22
<212> PRT
<213> Clostridium aminophilum
<400> 1162
Leu Arg Lys Ala Ile Tyr Ser Leu Arg Asn Glu Thr Phe His Phe Thr
1 5 10 15
Thr Leu Asn Lys Gly Ser
20
<210> 1163
<211> 22
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(22)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1163
Ile Ile Gln Ile Ile Tyr Ser Leu Arg Asn Lys Ser Phe His Phe Lys
1 5 10 15
Thr Tyr Asp His Gly Asp
20
<210> 1164
<211> 22
<212> PRT
<213> Carnobacterium gallinarum
<400> 1164
Leu Arg Gly Ser Val Gln Gln Ile Arg Asn Glu Ile Phe His Ser Phe
1 5 10 15
Asp Lys Asn Gln Lys Phe
20
<210> 1165
<211> 21
<212> PRT
<213> Carnobacterium gallinarum
<400> 1165
Ile Arg Gly Ala Val Gln Arg Val Arg Asn Gln Ile Phe His Gln Gln
1 5 10 15
Ile Asn Lys Arg His
20
<210> 1166
<211> 20
<212> PRT
<213> Paludibacter propionicigenes
<400> 1166
Ile Arg Gly Ala Val Gln Gln Ile Arg Asn Asn Val Asn His Tyr Lys
1 5 10 15
Lys Asp Ala Leu
20
<210> 1167
<211> 20
<212> PRT
<213> Listeria seeligeri
<400> 1167
Leu Arg Gly Ala Ile Ala Pro Ile Arg Asn Glu Ile Ile His Leu Lys
1 5 10 15
Lys His Ser Trp
20
<210> 1168
<211> 20
<212> PRT
<213> Listeria weihenstephanensis
<400> 1168
Ile Arg Gly Ser Ile Gln Gln Ile Arg Asn Glu Val Tyr His Cys Lys
1 5 10 15
Lys His Ser Trp
20
<210> 1169
<211> 20
<212> PRT
<213> Listeria newyorkensis
<400> 1169
Ile Arg Gly Ser Ile Gln Gln Ile Arg Asn Glu Val Tyr His Cys Lys
1 5 10 15
Lys His Ser Trp
20
<210> 1170
<211> 21
<212> PRT
<213> Leptotrichia wadei
<400> 1170
Ile Ser Tyr Ser Ile Tyr Asn Val Arg Asn Gly Val Gly His Phe Asn
1 5 10 15
Lys Leu Ile Leu Gly
20
<210> 1171
<211> 21
<212> PRT
<213> Leptotrichia wadei
<400> 1171
Met Leu Asn Ala Ile Thr Ser Ile Arg His Arg Val Val His Tyr Asn
1 5 10 15
Met Asn Thr Asn Ser
20
<210> 1172
<211> 21
<212> PRT
<213> Leptotrichia wadei
<400> 1172
Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe Asn
1 5 10 15
Leu Glu Leu Glu Gly
20
<210> 1173
<211> 22
<212> PRT
<213> Rhodobacter capsulatus
<400> 1173
Leu Leu Arg Tyr Leu Arg Gly Cys Arg Asn Gln Thr Phe His Leu Gly
1 5 10 15
Ala Arg Ala Gly Phe Leu
20
<210> 1174
<211> 21
<212> PRT
<213> Leptotrichia buccalis
<400> 1174
Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe Asn
1 5 10 15
Leu Glu Leu Glu Gly
20
<210> 1175
<211> 21
<212> PRT
<213> Leptotrichia sp.
<400> 1175
Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe Asn
1 5 10 15
Leu Glu Leu Glu Gly
20
<210> 1176
<211> 16
<212> PRT
<213> Leptotrichia sp.
<400> 1176
Phe Gln Lys Glu Gly Tyr Leu Leu Arg Asn Lys Ile Leu His Asn Ser
1 5 10 15
<210> 1177
<211> 15
<212> PRT
<213> Leptotrichia shahii
<400> 1177
Phe Thr Lys Ile Gly Thr Asn Glu Arg Asn Arg Ile Leu His Ala
1 5 10 15
<210> 1178
<211> 14
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1178
Phe Arg Asn Glu Ile Asp His Phe His Tyr Phe Tyr Asp Arg
1 5 10
<210> 1179
<211> 14
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1179
Leu Arg Asn Tyr Ile Glu His Phe Arg Tyr Tyr Ser Ser Phe
1 5 10
<210> 1180
<211> 14
<212> PRT
<213> Clostridium aminophilum
<400> 1180
Val Arg Lys Tyr Val Asp His Phe Lys Tyr Tyr Ala Thr Ser
1 5 10
<210> 1181
<211> 14
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1181
Leu Arg Lys Tyr Val Asp His Phe Lys Tyr Tyr Ala Tyr Gly
1 5 10
<210> 1182
<211> 14
<212> PRT
<213> Carnobacterium gallinarum
<400> 1182
Ile Arg Asn Gln Thr Ala His Leu Ser Val Leu Gln Leu Glu
1 5 10
<210> 1183
<211> 14
<212> PRT
<213> Carnobacterium gallinarum
<400> 1183
Ile Arg Asn Asn Ile Ala His Leu His Val Leu Arg Asn Asp
1 5 10
<210> 1184
<211> 14
<212> PRT
<213> Paludibacter propionicigenes
<400> 1184
Ile Arg Asn His Ile Ala His Phe Asn Tyr Leu Thr Lys Asp
1 5 10
<210> 1185
<211> 14
<212> PRT
<213> Listeria seeligeri
<400> 1185
Lys Arg Asn Asn Ile Ser His Phe Asn Tyr Leu Asn Gly Gln
1 5 10
<210> 1186
<211> 14
<212> PRT
<213> Listeria weihenstephanensis
<400> 1186
Ala Arg Asn His Ile Ala His Leu Asn Tyr Leu Ser Leu Lys
1 5 10
<210> 1187
<211> 14
<212> PRT
<213> Listeria newyorkensis
<400> 1187
Ala Arg Asn His Ile Ala His Leu Asn Tyr Leu Ser Leu Lys
1 5 10
<210> 1188
<211> 14
<212> PRT
<213> Leptotrichia wadei
<400> 1188
Phe Arg Asn Tyr Ile Ala His Phe Leu His Leu His Thr Lys
1 5 10
<210> 1189
<211> 14
<212> PRT
<213> Leptotrichia wadei
<400> 1189
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro Asp Ala
1 5 10
<210> 1190
<211> 14
<212> PRT
<213> Leptotrichia wadei
<400> 1190
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro His Ala
1 5 10
<210> 1191
<211> 14
<212> PRT
<213> Rhodobacter capsulatus
<400> 1191
Thr Arg Lys Asp Leu Ala His Phe Asn Val Leu Asp Arg Ala
1 5 10
<210> 1192
<211> 14
<212> PRT
<213> Leptotrichia buccalis
<400> 1192
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro His Ala
1 5 10
<210> 1193
<211> 14
<212> PRT
<213> Leptotrichia sp.
<400> 1193
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro Asn Ala
1 5 10
<210> 1194
<211> 14
<212> PRT
<213> Leptotrichia sp.
<400> 1194
Ile Arg Asn Tyr Ile Ser His Phe Tyr Ile Val Arg Asn Pro
1 5 10
<210> 1195
<211> 14
<212> PRT
<213> Leptotrichia shahii
<400> 1195
Ile Arg Asn Tyr Ile Ser His Phe Tyr Ile Val Arg Asn Pro
1 5 10
<210> 1196
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1196
gugccacuuc ucagaucgcu cgcucaguga uccgac 36
<210> 1197
<211> 105
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1197
gucagaacac ugagcgagcg uucuuuuuga gaagcucaac gggcuuugcc accuggaaag 60
uggccauugg cacacccguu gaaaaaauuc uguccucuag acaga 105
<210> 1198
<211> 105
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1198
gucagaacac ugagcgagcg uucuuuuuga gaagcucaac gggcuuugcc accuggaaag 60
uggccauugg cacacccguu gaaaaaauuc uguccucuag acaga 105
<210> 1199
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1199
gugccacuuc ucagaucgcu cgcucaguga uccgac 36
<210> 1200
<211> 37
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1200
gugccaauca cccaacacug accaagcuug ccgagac 37
<210> 1201
<211> 64
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1201
cuuggggaaa gcuaggcaag uuuuggauga uaagaaauaa ucaugucaca aggagggagu 60
uuuu 64
<210> 1202
<211> 64
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1202
cuuggggaaa gcuaggcaag uuuuggauga uaagaaauaa ucaugucaca aggagggagu 60
uuuu 64
<210> 1203
<211> 37
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1203
gugccaauca cccaacacug accaagcuug ccgagac 37
<210> 1204
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1204
gccgcagcga augccguuuc acgaaucguc aggcgg 36
<210> 1205
<211> 75
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1205
gcuggagacg uuuuuugaaa cggcgagugc ugcggauagc gaguuucucu uggggaggcg 60
cucgcggcca cuuuu 75
<210> 1206
<211> 75
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1206
gcuggagacg uuuuuugaaa cggcgagugc ugcggauagc gaguuucucu uggggaggcg 60
cucgcggcca cuuuu 75
<210> 1207
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1207
gccgcagcga augccguuuc acgaaucguc aggcgg 36
<210> 1208
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1208
guccaagaaa aaagaaauga uacgaggcau uagcac 36
<210> 1209
<211> 107
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1209
cuggacgaug ucucuuuuau uucuuuuuuc uuggaucuga guacgagcac ccacauugga 60
cauuucgcau ggugggugcu cguacuauag guaaaacaaa ccuuuuu 107
<210> 1210
<211> 107
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1210
cuggacgaug ucucuuuuau uucuuuuuuc uuggaucuga guacgagcac ccacauugga 60
cauuucgcau ggugggugcu cguacuauag guaaaacaaa ccuuuuu 107
<210> 1211
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1211
guccaagaaa aaagaaauga uacgaggcau uagcac 36
<210> 1212
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1212
guucgaaagc uuaguggaaa gcuucguccu uagcac 36
<210> 1213
<211> 69
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1213
cacggauaau cacgacuuuc cacuaagcuu ucgaauuuua ugaugcgagc auccucucag 60
gucaaaaaa 69
<210> 1214
<211> 69
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1214
cacggauaau cacgacuuuc cacuaagcuu ucgaauuuua ugaugcgagc auccucucag 60
gucaaaaaa 69
<210> 1215
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1215
guucgaaagc uuaguggaaa gcuucguggu uagcac 36
<210> 1216
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1216
guauugagaa aagccagaua uaguuggcaa uagac 35
<210> 1217
<211> 62
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1217
auauuuugau ucccauuuau gguuauuuac cauaaauggg aaucaacuaa aaaauauuuu 60
uu 62
<210> 1218
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1218
guauugagaa aagccagaua uaguuggcaa uagac 35
<210> 1219
<211> 62
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1219
auauuuugau ucccauuuau gguuauuuac cauaaauggg aaucaacuaa aaaauauuuu 60
uu 62
<210> 1220
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1220
guugaugaga agagcccaag auagagggca auaac 35
<210> 1221
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1221
gcuggagaag auagcccaag aaagagggca auaac 35
<210> 1222
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1222
auuauuacca uuuugguugg aaugcuauua uaaaggauca uucgauuauu accucuaccu 60
cccuucccac gauuucuu 78
<210> 1223
<211> 78
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1223
attattacca ttttggttgg aatgctatta taaaggatca ttcgattatt acctctacct 60
cccttcccac gatttctt 78
<210> 1224
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1224
guugaugaga agagcccaag auagagggca auaac 35
<210> 1225
<211> 78
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1225
attattacca ttttggttgg aatgctatta taaaggatca ttcgattatt acctctacct 60
cccttcccac gatttctt 78
<210> 1226
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1226
gcuggagaag auagcccaag aaagagggca auaac 35
<210> 1227
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1227
guuuggagaa cagcccgaua uagagggcaa uagac 35
<210> 1228
<211> 81
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1228
gucuuacgac cucaguauua ggaagauuuc aaccaagaaa acuuaguuuc aggcuuaaug 60
aucgagucau gcagccaaag u 81
<210> 1229
<211> 81
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1229
gucuuacgac cucaguauua ggaagauuuc aaccaagaaa acuuaguuuc aggcuuaaug 60
aucgagucau gcagccaaag u 81
<210> 1230
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1230
guuuggagaa cagcccgaua uagagggcaa uagac 35
<210> 1231
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1231
guuuugagaa uagcccgaca uagagggcaa uagac 35
<210> 1232
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1232
guuaugaaaa cagcccgaca uagagggcaa uagaca 36
<210> 1233
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1233
guuauagucc ucuuacauuu agagguaguc uuuaau 36
<210> 1234
<211> 98
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1234
ucuuaagaac uucucuaccu gaaguuggau uauaaaugac ucuugcucuc auagauaucc 60
uccuuugaaa auauacacug ccgauuaauu accguuuu 98
<210> 1235
<211> 98
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1235
ucuuaagaac uucucuaccu gaaguuggau uauaaaugac ucuugcucuc auagauaucc 60
uccuuugaaa auauacacug ccgauuaauu accguuuu 98
<210> 1236
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1236
guuauagucc ucuuacauuu agagguaguc uuuaau 36
<210> 1237
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1237
guuauagucc ucuuacauuu agagguaguu uauauu 36
<210> 1238
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1238
guuauagucc ccuuacauuu agggguaguc uuuaau 36
<210> 1239
<211> 102
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1239
aauauaaauu cucccuaaau auaagagaau aauaacucaa ucucuucauu cguauuuugu 60
cuaguuaaga uaaguaccac caaauacaau caauccaaaa aa 102
<210> 1240
<211> 102
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1240
aauauaaauu cucccuaaau auaagagaau aauaacucaa ucucuucauu cguauuuugu 60
cuaguuaaga uaaguaccac caaauacaau caauccaaaa aa 102
<210> 1241
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1241
guuauagucc ucuuacauuu agagguaguu uauauu 36
<210> 1242
<211> 102
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1242
aauauaaauu cucccuaaau auaagagaau aauaacucaa ucucuucauu cguauuuugu 60
cuaguuaaga uaaguaccac caaauacaau caauccaaaa aa 102
<210> 1243
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1243
guuauagucc ccuuacauuu agggguaguc uuuaau 36
<210> 1244
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1244
guuguaguuc ccuucaauuu ugggauaauc cacaag 36
<210> 1245
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1245
guuuuagucc ucuuucauau agagguaguc ucuuac 36
<210> 1246
<211> 99
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1246
augaaaagag gacuaaaacu gaaagaggac uaaaacacca gauguggaua acuauauuag 60
uggcuauuaa aaauucgucg auauuagaga ggaaacuuu 99
<210> 1247
<211> 99
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1247
augaaaagag gacuaaaacu gaaagaggac uaaaacacca gauguggaua acuauauuag 60
uggcuauuaa aaauucgucg auauuagaga ggaaacuuu 99
<210> 1248
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1248
guuuuagucc ucuuucauau agagguaguc ucuuac 36
<210> 1249
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1249
guuuuagacc ucuucuauuu ugagguacuc uaaauc 36
<210> 1250
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1250
guuuuagucc ucuuuuguuu ugagguacuc uaaauc 36
<210> 1251
<211> 147
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1251
aagucagcgc acaacaaaga agaugacgaa caaaaucucu cgccaucuuc uuaaaauuau 60
uugccacaca gccaacauua uaagcguuaa aaccagcacc augaguacau uucacccaac 120
aaucagaauc cccguuucuc cguuuuu 147
<210> 1252
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1252
guuuuagucc ucuuuuguuu ugagguacuc uaaauc 36
<210> 1253
<211> 147
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1253
aagucagcgc acaacaaaga agaugacgaa caaaaucucu cgccaucuuc uuaaaauuau 60
uugccacaca gccaacauua uaagcguuaa aaccagcacc augaguacau uucacccaac 120
aaucagaauc cccguuucuc cguuuuu 147
<210> 1254
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1254
guuuuagauc ccuucguuuu ugggguuauc uauauc 36
<210> 1255
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1255
guuuuagucc ccuucguuuu ugggguaguc uaaauc 36
<210> 1256
<211> 113
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1256
gauuuagagc accccaaaag uaaugaaaau uugcaauuaa auaaggaaua uuaaaaaaau 60
gugauuuuaa aaaaauugaa gaaauuaaau gaaaaauugu ccaaguaaaa aaa 113
<210> 1257
<211> 70
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1257
auuuagauua ccccuuuaau uuauuuuacc auauuuuucu cauaaugcaa acuaauauuc 60
caaaauuuuu 70
<210> 1258
<211> 113
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1258
gauuuagagc accccaaaag uaaugaaaau uugcaauuaa auaaggaaua uuaaaaaaau 60
gugauuuuaa aaaaauugaa gaaauuaaau gaaaaauugu ccaaguaaaa aaa 113
<210> 1259
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1259
guuuuagucc ccuucguuuu ugggguaguc uaaauc 36
<210> 1260
<211> 70
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1260
auuuagauua ccccuuuaau uuauuuuacc auauuuuucu cauaaugcaa acuaauauuc 60
caaaauuuuu 70
<210> 1261
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1261
guuuuagucc ccuucguuuu ugggguaguc uaaauc 36
<210> 1262
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1262
guuuuagucc ccuucgauau uggggugguc uauauc 36
<210> 1263
<211> 95
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1263
auugaugugg uauacuaaaa auggaaaauu guauuuuuga uuagaaagau guaaaauuga 60
uuuaauuuaa aaauauuuua uuagauuaaa guaga 95
<210> 1264
<211> 95
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1264
auugaugugg uauacuaaaa auggaaaauu guauuuuuga uuagaaagau guaaaauuga 60
uuuaauuuaa aaauauuuua uuagauuaaa guaga 95
<210> 1265
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1265
guuuuagucc ccuucgauau uggggugguc uauauc 36
<210> 1266
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1266
guucaguccg ccgucgucuu ggcggugaug ugaggc 36
<210> 1267
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1267
guucaguccg ccgucauuuu ggcggugaug ugcucc 36
<210> 1268
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1268
guucaguccg ccgucgucuu ggcggugaug ugaggc 36
<210> 1269
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1269
guucaguccg ccgucauuuu ggcggugaug ugcucc 36
<210> 1270
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1270
guucaguccg ccgucgucuu ggcggugaug ugaggc 36
<210> 1271
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1271
guucaguccg ccgucauuuu ggcggugaug ugcucc 36
<210> 1272
<211> 81
<212> DNA
<213> Alicyclobacillus acidoterrestris
<400> 1272
agcgcccgct gtcggatcac tgagcgagcg atctgagaag tggcactgtt tggtaaaggt 60
aaaaagacga atgatgcatc c 81
<210> 1273
<211> 83
<212> DNA
<213> Alicyclobacillus acidoterrestris
<400> 1273
tgatgcatcc gtcggatcac tgagcgagcg atctgagaag tggcaccctt tataaaaagg 60
ggcgtccttt agtaccgtgt act 83
<210> 1274
<211> 84
<212> DNA
<213> Alicyclobacillus acidoterrestris
<400> 1274
accgtgtact gtcggatcac tgagcgagcg atctgagaag tggcacaagc cttgagtaat 60
tcgccgtggg attccccgcc gtat 84
<210> 1275
<211> 80
<212> DNA
<213> Alicyclobacillus acidoterrestris
<400> 1275
cccgccgtat gtcggatcac tgagcgagcg atctgagaag tggcactaat gaagttaaag 60
gagatgagac aatgaaagaa 80
<210> 1276
<211> 83
<212> DNA
<213> Alicyclobacillus acidoterrestris
<400> 1276
aatgaaagaa gtcggatcac tgagcgagcg atctgagaag tggcactgca atgcgttgga 60
ttatgacgat gcaggccaag gaa 83
<210> 1277
<211> 56
<212> DNA
<213> Alicyclobacillus acidoterrestris
<400> 1277
ggccaaggaa gtcggatcac tgagcgagcg atctgagaag tggcactgat gtcagc 56
<210> 1278
<211> 36
<212> DNA
<213> Alicyclobacillus acidoterrestris
<400> 1278
gtcggatcac tgagcgagcg atctgagaag tggcac 36
<210> 1279
<211> 36
<212> DNA
<213> Alicyclobacillus acidoterrestris
<400> 1279
gtcggatcac tgagcgagcg atctgagaag tggcac 36
<210> 1280
<211> 84
<212> DNA
<213> Desulfonatronum thiodismutans
<400> 1280
ccggctcgag gtctcggcaa gcttggtcag tgttgggtga ttggcacatc caggtggttg 60
gatgcgggac ataccttccg cctt 84
<210> 1281
<211> 84
<212> DNA
<213> Desulfonatronum thiodismutans
<400> 1281
cttccgcctt gtctcggcaa gcttggtcag tgttgggtga ttggcactgc ttcccggcga 60
acggcgagct gacctcctag atgt 84
<210> 1282
<211> 83
<212> DNA
<213> Desulfonatronum thiodismutans
<400> 1282
tcctagatgt gtctcggcaa gcttggtcag tgttgggtga ttggcaccgt ctgctcggtc 60
tcggacttca ccaccacgtc cac 83
<210> 1283
<211> 83
<212> DNA
<213> Desulfonatronum thiodismutans
<400> 1283
ccacgtccac gtctcggcaa gcttggtcag tgttgggtga ttggcacggg tgcggcattt 60
gcgggtgttg ggggagtggc agg 83
<210> 1284
<211> 37
<212> DNA
<213> Desulfonatronum thiodismutans
<400> 1284
gtctcggcaa gcttggtcag tgttgggtga ttggcac 37
<210> 1285
<211> 15
<212> DNA
<213> Desulfonatronum thiodismutans
<400> 1285
gtctcggcaa gcttg 15
<210> 1286
<211> 22
<212> DNA
<213> Desulfonatronum thiodismutans
<400> 1286
gtcagtgttg ggtgattggc ac 22
<210> 1287
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1287
acatccactg ccgcctgacg attcgtgaaa cggcattcgc tgcggcaata gtctctggaa 60
atgttatagt agctcctaca 80
<210> 1288
<211> 81
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(81)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1288
agctcctaca ccgcctgacg attcgtgaaa cggcattcgc tgcggcaaga aaaagagtcg 60
tggtgttggc gcgggtcaga c 81
<210> 1289
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1289
cgggtcagac ccgcctgacg attcgtgaaa cggcattcgc tgcggcaatg taacgcctgg 60
agcatggctt gacccgaacc 80
<210> 1290
<211> 82
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(82)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1290
gacccgaacc ccgcctgacg attcgtgaaa cggcattcgc tgcggcaata tacgtctgat 60
taaaggtatg ggattccctg tt 82
<210> 1291
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1291
attccctgtt ccgcctgacg attcgtgaaa cggcattcgc tgcggctctc agtcaattcg 60
aatatgatgc ggggtactgg 80
<210> 1292
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1292
ggggtactgg ccgcctgacg attcgtgaaa cggcattcgc tgcggcgctc cacaaaagcg 60
attatcattt cccggttata 80
<210> 1293
<211> 82
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(82)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1293
cccggttata ccgcctgacg attcgtgaaa cggcattcgc tgcggcgtgc ccggccatgc 60
ggttatcggt ctcgatggcc tt 82
<210> 1294
<211> 79
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(79)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1294
cgatggcctt ccgcctgacg attcgtgaaa cggcattcgc tgcggctcgc ggggaaacga 60
gtgcgtagtc gatcgtcac 79
<210> 1295
<211> 79
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(79)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1295
cgatcgtcac ccgcctgacg attcgtgaaa cggcattcgc tgcggcgtag ctgtcgccgt 60
ctttcttgta ttctttttt 79
<210> 1296
<211> 82
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(82)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1296
attctttttt ccgcctgacg attcgtgaaa cggcattcgc tgcggcgatc ggacaatcac 60
gccagacatt gccggtcatg at 82
<210> 1297
<211> 83
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(83)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1297
cggtcatgat ccgcctgacg attcgtgaaa cggcattcgc tgcggcttgg cgaccttcag 60
gcgagcgtta ttggcggcat aga 83
<210> 1298
<211> 81
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(81)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1298
gcggcataga ccgcctgacg attcgtgaaa cggcattcgc tgcggcaaaa atggcgaaac 60
cgaagccgcc gacgcgatac a 81
<210> 1299
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1299
acgcgataca ccgcctgacg attcgtgaaa cggcattcgc tgcggcatgt caattttggt 60
aacacttcgc cttggcacca 80
<210> 1300
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1300
cttggcacca ccgcctgacg attcgtgaaa cggcattcgc tgcggcatgt caattttggt 60
aacacttcgc cttggcacca 80
<210> 1301
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1301
cttggcacca ccgcctgacg attcgtgaaa cggcattcgc tgcggcagca cgtggggttt 60
ttgctctcac aaagtaaatt 80
<210> 1302
<211> 79
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(79)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1302
aaagtaaatt ccgcctgacg attcgtgaaa cggcattcgc tgcggcatag catcggcgag 60
tgtgtctgac agtcctact 79
<210> 1303
<211> 83
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(83)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1303
cagtcctact ccgcctgacg attcgtgaaa cggcattcgc tgcggcggcg aaccctcgtc 60
aaccgcgacc tcaagatggc aca 83
<210> 1304
<211> 81
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(81)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1304
agatggcaca ccgcctgacg attcgtgaaa cggcattcgc tgcggcaacg catcacgcgc 60
ctccggctcc atttccttgc c 81
<210> 1305
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1305
tttccttgcc ccgcctgacg attcgtgaaa cggcattcgc tgcggctccg ctcacagggg 60
caatctacgc tcaggagatg 80
<210> 1306
<211> 81
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(81)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1306
tcaggagatg ccgcctgacg attcgtgaaa cggcattcgc tgcggcgaaa acgccccgaa 60
acacagttcc cgaattcaaa t 81
<210> 1307
<211> 79
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(79)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1307
gaattcaaat ccgcctgacg attcgtgaaa cggcattcgc tgcggcagca caattccgag 60
aaacggtttc aggatcata 79
<210> 1308
<211> 81
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(81)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1308
caggatcata ccgcctgacg attcgtgaaa cggcattcgc tgcggcgcca gcgttccctg 60
cgatgctgtt gcgaaatccc c 81
<210> 1309
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1309
cgaaatcccc ccgcctgacg attcgtgaaa cggcattcgc tgcggcttgc actgcgtggt 60
gaaagagttc aaagcgtcca 80
<210> 1310
<211> 56
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(56)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1310
aaagcgtcca ccgcctgacg attcgtgaaa cggcattcgc tgcggcccgt gctcgc 56
<210> 1311
<211> 36
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(36)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1311
ccgcctgacg attcgtgaaa cggcattcgc tgcggc 36
<210> 1312
<211> 36
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(36)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1312
ccgcctgacg attcgtgaaa cggcattcgc tgcggc 36
<210> 1313
<211> 10
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(10)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1313
ccgcctgacg 10
<210> 1314
<211> 12
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(12)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1314
attcgtgaaa cg 12
<210> 1315
<211> 14
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1315
gcattcgctg cggc 14
<210> 1316
<211> 10
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(10)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1316
ccgcctgacg 10
<210> 1317
<211> 12
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(12)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1317
attcgtgaaa cg 12
<210> 1318
<211> 14
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1318
gcattcgctg cggc 14
<210> 1319
<211> 36
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(36)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1319
ccgcctgacg attcgtgaaa cggcattcgc tgcggc 36
<210> 1320
<211> 10
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(10)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1320
ccgcctgacg 10
<210> 1321
<211> 12
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(12)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1321
attcgtgaaa cg 12
<210> 1322
<211> 14
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1322
gcattcgctg cggc 14
<210> 1323
<211> 36
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(36)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1323
ccgcctgacg attcgtgaaa cggcattcgc tgcggc 36
<210> 1324
<211> 10
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(10)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1324
ccgcctgacg 10
<210> 1325
<211> 12
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(12)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1325
attcgtgaaa cg 12
<210> 1326
<211> 14
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1326
gcattcgctg cggc 14
<210> 1327
<211> 81
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1327
accccctgcg gtccaagaaa aaagaaatga tacgaggcat tagcaccatg caaacggatt 60
gttatataaa tcttcttgaa c 81
<210> 1328
<211> 82
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1328
cttcttgaac gtccaagaaa aaagaaatga tacgaggcat tagcacattg ttccggcggc 60
taatttgtct gcggtaatcg aa 82
<210> 1329
<211> 87
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1329
ggtaatcgaa gtccaagaaa aaagaaatga tacgaggcat tagcaccttc gccatcctca 60
tcccttatca gttgattgcc tagcgtt 87
<210> 1330
<211> 83
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1330
gcctagcgtt gtccaagaaa aaagaaatga tacgaggcat tagcacacac agaaaccaaa 60
tgggaacacg ttttcgttaa taa 83
<210> 1331
<211> 84
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1331
tcgttaataa gtccaagaaa aaagaaatga tacgaggcat tagcacgaca ttaaaaaatt 60
ccaaccaagc gagttattga gtgg 84
<210> 1332
<211> 80
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1332
tattgagtgg gtccaagaaa aaagaaatga tacgaggcat tagcacttat gagcttaaaa 60
gcttgttagc gacattaaac 80
<210> 1333
<211> 83
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1333
gacattaaac gtccaagaaa aaagaaatga tacgaggcat tagcacgttt tgttacagct 60
ttattcctta cttgatcgac tct 83
<210> 1334
<211> 83
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1334
gatcgactct gtccaagaaa aaagaaatga tacgaggcat tagcaccaga acctatctca 60
agaggatgca ttctggaaag gaa 83
<210> 1335
<211> 86
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1335
tggaaaggaa gtccaagaaa aaagaaatga tacgaggcat tagcacctta taacaataat 60
ttaaaagcaa tttatgactg tataga 86
<210> 1336
<211> 82
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1336
actgtataga gtccaagaaa aaagaaatga tacgaggcat tagcactttt aagggacatc 60
agaaacacta taagctcact tg 82
<210> 1337
<211> 81
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1337
agctcacttg gtccaagaaa aaagaaatga tacgaggcat tagcacataa tcgactttgc 60
atttctatag tgtcgttcat c 81
<210> 1338
<211> 85
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1338
gtcgttcatc gtccaagaaa aaagaaatga tacgaggcat tagcacaaaa tggaacaagg 60
aacaatagac gtttataagt atgga 85
<210> 1339
<211> 83
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1339
taagtatgga gtccaagaaa aaagaaatga tacgaggcat tagcactttc aataaagcat 60
caaactcttt ttgcattttt tca 83
<210> 1340
<211> 83
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1340
cattttttca gtccaagaaa aaagaaatga tacgaggcat tagcacaccg agtaaggaat 60
cgtttaatca acaaacttga aac 83
<210> 1341
<211> 84
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1341
aacttgaaac gtccaagaaa aaagaaatga tacgaggcat tagcacggta gtcggcggct 60
aagtgtcgca gggttaacac cgat 84
<210> 1342
<211> 85
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1342
taacaccgat gtccaagaaa aaagaaatga tacgaggcat tagcacacgc tgaacaaaac 60
tcacgaaacc aaaagtttat aaaat 85
<210> 1343
<211> 81
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1343
tttataaaat gtccaagaaa aaagaaatga tacgaggcat tagcacggaa aggatttact 60
agatctcgca agaaaggtaa c 81
<210> 1344
<211> 82
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1344
gaaaggtaac gtccaagaaa aaagaaatga tacgaggcat tagcacattt agtatattgt 60
tgttttcatt tgcttttttc gc 82
<210> 1345
<211> 83
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1345
cttttttcgc gtccaagaaa aaagaaatga tacgaggcat tagcacttat accgtaaaaa 60
attttggatt tgatgtcacc gtc 83
<210> 1346
<211> 82
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1346
tgtcaccgtc gtccaagaaa aaagaaatga tacgaggcat tagcacagaa cacaaaaagc 60
ggaaaaattg cacttatttt cg 82
<210> 1347
<211> 82
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1347
cttattttcg gtccaagaaa aaagaaatga tacgaggcat tagcacaata ctcgtctaca 60
aactttttct gcttttctgt ta 82
<210> 1348
<211> 84
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1348
ttttctgtta gtccaagaaa aaagaaatga tacgaggcat tagcaccata caggacactt 60
aaactctact ttacgatttt caaa 84
<210> 1349
<211> 82
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1349
gattttcaaa gtccaagaaa aaagaaatga tatgaggcat tagcacgatt taaaacttct 60
tctggcatcc agtacataga tt 82
<210> 1350
<211> 82
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1350
tacatagatt gtccaagaaa aaagaaatga tatgaggcat tagcacgaaa aaactgtttc 60
cttatcacac ctatagcaat aa 82
<210> 1351
<211> 84
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1351
atagcaataa gtccaagaaa aaagaaatga tatgaggcat tagcactcta gatatgttca 60
agaaaatatg cagtctatcg gtca 84
<210> 1352
<211> 85
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1352
ctatcggtca gtccaagaaa aaagaaatga tatgaggcat tagcacgaat caattaatcc 60
aatttgttgc aacttaggta gagag 85
<210> 1353
<211> 84
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1353
aggtagagag gtccaagaaa aaagaaatga tatgaggcat tagcacccga attagcatcg 60
taccaattac atccatatga tgct 84
<210> 1354
<211> 82
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1354
atatgatgct gtccaagaaa aaagaaatga tatgaggcat tagcactttt ctatttttat 60
attactgttt gtttggtgat aa 82
<210> 1355
<211> 84
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1355
ttggtgataa gtccaagaaa aaagaaatga tatgaggcat tagcactgaa tctaaccaag 60
gaagaaataa aagatattta taag 84
<210> 1356
<211> 81
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1356
tatttataag gtccaagaaa aaagaaatga tatgaggcat tagcacaaaa aggagtgttt 60
caaaatggca aatgaaaaaa g 81
<210> 1357
<211> 82
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1357
atgaaaaaag gtccaagaaa aaagaaatga tatgaggcat tagcacagct tggattgaat 60
caaacaatgg tggtcgaggt tt 82
<210> 1358
<211> 82
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1358
gtcgaggttt gtccaagaaa aaagaaatga tatgaggcat tagcacttga gttgtaatta 60
atcaccgttc tacctgaaaa ct 82
<210> 1359
<211> 80
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1359
cctgaaaact gtccaagaaa aaagaaatga tatgaggcat tagcactaga tgttgaaaac 60
ggcggttttg tctattatcc 80
<210> 1360
<211> 84
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1360
tctattatcc gtccaagaaa aaagaaatga tatgaggcat tagcacaaac ccgcaaaaaa 60
taagctgttg acgagtctag ttag 84
<210> 1361
<211> 85
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1361
gtctagttag gtccaagaaa aaagaaatga tacgaggcat tagcacttgt caatcctcga 60
aaatgccggg cggacgggct tgcca 85
<210> 1362
<211> 81
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1362
gggcttgcca gtccaagaaa aaagaaatga aacgaggcat tagcacccac caccgacggc 60
gtccacgcca attgcctgct t 81
<210> 1363
<211> 72
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1363
ttgcctgctt gtccaagaaa aaagaaatga tacgaggcat tagcacaaca atataaacga 60
ctactttacc gt 72
<210> 1364
<211> 56
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1364
actttaccgt gttcaagaaa aaagaaatga tatgaggcat tagcacgatg ggatgg 56
<210> 1365
<211> 36
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1365
gtccaagaaa aaagaaatga tacgaggcat tagcac 36
<210> 1366
<211> 36
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1366
gtccaagaaa aaagaaatga tacgaggcat tagcac 36
<210> 1367
<211> 36
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1367
gtccaagaaa aaagaaatga tacgaggcat tagcac 36
<210> 1368
<211> 83
<212> DNA
<213> Bacillus sp.
<400> 1368
ggactttagc gtgctaacca cgaagctttc cactaagctt tcgaacaccg gattggtgga 60
aagacgccgc tgccgaccga aaa 83
<210> 1369
<211> 83
<212> DNA
<213> Bacillus sp.
<400> 1369
cgaccgaaaa gtgctaacca cgaagctttc cactaagctt tcgaacaccg gattggtgga 60
aagacgccgc tgccgaccga aaa 83
<210> 1370
<211> 87
<212> DNA
<213> Bacillus sp.
<400> 1370
cgaccgaaaa gtgctaacca cgaagctttc cactaagctt tcgaacggga aaacgtgggc 60
gcaactttgc tattctggca tgtttgc 87
<210> 1371
<211> 83
<212> DNA
<213> Bacillus sp.
<400> 1371
gcatgtttgc gtgctaacca cgaagctttc cactaagctt tcgaacaagt ttggaagggg 60
gcgatagcga tgggttttaa gat 83
<210> 1372
<211> 81
<212> DNA
<213> Bacillus sp.
<400> 1372
gttttaagat gtgctaacca cgaagctttc cactaagctt tcgaacccga tcaccgtcaa 60
aaccgtagta gtaagactcg c 81
<210> 1373
<211> 81
<212> DNA
<213> Bacillus sp.
<400> 1373
taagactcgc gtgctaacca cgaagctttc cactaagctt tcgaacaagg cggcctaaat 60
cacttgggcc gcccttaaga t 81
<210> 1374
<211> 86
<212> DNA
<213> Bacillus sp.
<400> 1374
cccttaagat gtgctaacca cgaagctttc cactaagctt tcgaactgct tttatttcca 60
tgctaccatt ggctgaatcg ggactg 86
<210> 1375
<211> 82
<212> DNA
<213> Bacillus sp.
<400> 1375
atcgggactg gtgctaacca cgaagctttc cactaagctt tcgaacaaac aggtatctgc 60
ttatcaacgt gcagcacagg cg 82
<210> 1376
<211> 82
<212> DNA
<213> Bacillus sp.
<400> 1376
agcacaggcg gtgctaacca cgaagctttc cactaagctt tcgaacctat atgagtagaa 60
cgtctctcaa taagcgtaga at 82
<210> 1377
<211> 81
<212> DNA
<213> Bacillus sp.
<400> 1377
agcgtagaat gtgctaacca cgaagctttc cactaagctt tcgaacgcgc cttgttgcta 60
tcattagggt cgcgatcaac a 81
<210> 1378
<211> 88
<212> DNA
<213> Bacillus sp.
<400> 1378
gcgatcaaca gtgctaacca cgaagctttc cactaagctt tcgaacgtat aacttttaga 60
taacaaatgt tatacaaaat gcttgacg 88
<210> 1379
<211> 81
<212> DNA
<213> Bacillus sp.
<400> 1379
atgcttgacg gtgctaacca cgaagctttc cactaagctt tcgaacagcc caagctttac 60
atacacctat gcgtatgctt t 81
<210> 1380
<211> 56
<212> DNA
<213> Bacillus sp.
<400> 1380
cgtatgcttt gtgctaacca cgaagctttc cactaagctt tcgaactcct tcccac 56
<210> 1381
<211> 36
<212> DNA
<213> Bacillus sp.
<400> 1381
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1382
<211> 84
<212> DNA
<213> Bacillus sp.
<400> 1382
tggacaagct gtgctaacca cgaagctttc cactaagctt tcgaacctct ggtgtaaggc 60
ggtttcttgc ttgtaagtgc gcgg 84
<210> 1383
<211> 84
<212> DNA
<213> Bacillus sp.
<400> 1383
aagtgcgcgg gtgctaatcc cgaagctttc cactaagctt tcgaacaaaa gagtataagt 60
accactctta gccgagtagt tcaa 84
<210> 1384
<211> 80
<212> DNA
<213> Bacillus sp.
<400> 1384
agtagttcaa gtgctaatcc cgaagctttc cactaagctt tcgaacactg gagaaccttc 60
caagtgtgcg atagcgtatc 80
<210> 1385
<211> 80
<212> DNA
<213> Bacillus sp.
<400> 1385
atagcgtatc gtgctaatcc cgaagctttc cactaagctt tcgaactttt ttgttccgcc 60
aaggtaaaac gtaccgagtt 80
<210> 1386
<211> 83
<212> DNA
<213> Bacillus sp.
<400> 1386
gtaccgagtt gtgctaatcc cgaagctttc cactaagctt tcgaactttc cgtaaaactt 60
tttgagaaaa caggagggcg act 83
<210> 1387
<211> 86
<212> DNA
<213> Bacillus sp.
<400> 1387
gagggcgact gtgctaatcc cgaagctttc cactaagctt tcgaaccagg ctttttgtgc 60
ataggtcatg cagtaaccta atccgc 86
<210> 1388
<211> 85
<212> DNA
<213> Bacillus sp.
<400> 1388
cctaatccgc gtgctaatcc cgaagctttc cactaagctt tcgaacgacc atgatctcga 60
aaagaaaatt acgcgtgcat cgcaa 85
<210> 1389
<211> 84
<212> DNA
<213> Bacillus sp.
<400> 1389
tgcatcgcaa gtgctaatcc cgaagctttc cactaagctt tcgaactggg ccgctccgat 60
tcgataaaga cgggtatctg gaga 84
<210> 1390
<211> 56
<212> DNA
<213> Bacillus sp.
<400> 1390
tatctggaga gtgctaatcc cgaagctttc cactaagctt tcgaaccagg gtaccc 56
<210> 1391
<211> 36
<212> DNA
<213> Bacillus sp.
<400> 1391
gtgctaatcc cgaagctttc cactaagctt tcgaac 36
<210> 1392
<211> 82
<212> DNA
<213> Bacillus sp.
<400> 1392
gctacctctg gtacatcccc ttcaatttcc actaagcttt cgaacgttcg gcgtagtagt 60
gtacacgctc ttgaatatga tg 82
<210> 1393
<211> 81
<212> DNA
<213> Bacillus sp.
<400> 1393
gaatatgatg gtacatcccc ttcaatttcc actaagcttt cgaacgccgc aacgtgctcc 60
gggagaggcg cacgttcaga g 81
<210> 1394
<211> 80
<212> DNA
<213> Bacillus sp.
<400> 1394
acgttcagag gtacatcccc ttcaatttcc actaagcttt cgaacgcgac cagtccgagc 60
agggcaatcc cgacaatgcg 80
<210> 1395
<211> 81
<212> DNA
<213> Bacillus sp.
<400> 1395
cgacaatgcg gtacatcccc ttcaatttcc actaagcttt cgaacaaccc gaaggactac 60
accatttccc gtgataaagc g 81
<210> 1396
<211> 81
<212> DNA
<213> Bacillus sp.
<400> 1396
tgataaagcg gtacatcccc ttcaatttcc actaagcttt cgaacaaaag cttggggagg 60
gcttcgagtc gggtatacag g 81
<210> 1397
<211> 79
<212> DNA
<213> Bacillus sp.
<400> 1397
ggtatacagg gtacatcccc ttcaatttcc actaagcttt cgaacccttg tccgcgataa 60
atctgtacgt cgaatccgt 79
<210> 1398
<211> 81
<212> DNA
<213> Bacillus sp.
<400> 1398
tcgaatccgt gtacatcccc ttcaatttcc actaagcttt cgaactgttt ggttgctgcg 60
cggccgaagt tgtctgacag t 81
<210> 1399
<211> 84
<212> DNA
<213> Bacillus sp.
<400> 1399
gtctgacagt gtacatcccc ttcaatttcc actaagcttt cgaacccgtt attaagttac 60
ctctttctag cataacctga gtgt 84
<210> 1400
<211> 88
<212> DNA
<213> Bacillus sp.
<400> 1400
acctgagtgt gtacatcccc ttcaatttcc actaagcttt cgaacatgta catcctgcat 60
cagttcgaag gaagcgggat cagctagt 88
<210> 1401
<211> 86
<212> DNA
<213> Bacillus sp.
<400> 1401
atcagctagt gtacatcccc ttcaatttcc actaagcttt cgaaccaaga gacgtgttga 60
cgatcatctt ttttgagatc tgccgt 86
<210> 1402
<211> 87
<212> DNA
<213> Bacillus sp.
<400> 1402
gatctgccgt gtacatcccc ttcaatttcc actaagcttt cgaacgcgca actcttgcaa 60
agttttttgc agcaacattt tttccgt 87
<210> 1403
<211> 82
<212> DNA
<213> Bacillus sp.
<400> 1403
ttttttccgt gtacatcccc ttcaatttcc actaagcttt cgaactcaac catacttatt 60
tattttagca ctgccccaaa gt 82
<210> 1404
<211> 56
<212> DNA
<213> Bacillus sp.
<400> 1404
gccccaaagt gtacatcccc ttcaatttcc actaagcttt cgaacaacct ctgctg 56
<210> 1405
<211> 35
<212> DNA
<213> Bacillus sp.
<400> 1405
gtacatcccc ttcaatttcc actaagcttt cgaac 35
<210> 1406
<211> 36
<212> DNA
<213> Bacillus sp.
<400> 1406
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1407
<211> 83
<212> DNA
<213> Desulfatirhabdium butyrativorans
<400> 1407
cccatccatt tgtccgtcga tcaagctgct ttcaccatcg gaaccccggc caccgaggca 60
gccaacacca cccaggccga gct 83
<210> 1408
<211> 83
<212> DNA
<213> Desulfatirhabdium butyrativorans
<400> 1408
aggccgagct gtgtcagtcg atcaagctgt tttcaccatc ggaacccctt gaattgagac 60
ggttccggag cgcaatatgg tga 83
<210> 1409
<211> 59
<212> DNA
<213> Desulfatirhabdium butyrativorans
<400> 1409
aatatggtga gtgtcagtcg atcaagctgt tttcaccatc ggaacccccc ccggccatc 59
<210> 1410
<211> 38
<212> DNA
<213> Desulfatirhabdium butyrativorans
<400> 1410
gtgtcagtcg atcaagctgt tttcaccatc ggaacccc 38
<210> 1411
<211> 38
<212> DNA
<213> Desulfatirhabdium butyrativorans
<400> 1411
gtgtcagtcg atcaagctgt tttcaccatc ggaacccc 38
<210> 1412
<211> 19
<212> DNA
<213> Desulfatirhabdium butyrativorans
<400> 1412
gtgtcagtcg atcaagctg 19
<210> 1413
<211> 19
<212> DNA
<213> Desulfatirhabdium butyrativorans
<400> 1413
ttttcaccat cggaacccc 19
<210> 1414
<211> 82
<212> DNA
<213> Brevibacillus agri
<400> 1414
cgaggatttt gtgctaacca cgaagctttc cactaagctt tcgaaccata ccgccgttta 60
ctttacctgg tacggcgact at 82
<210> 1415
<211> 81
<212> DNA
<213> Brevibacillus agri
<400> 1415
cggcgactat gtgctaacca cgaagctttc cactaagctt tcgaactctg caagtgcgcg 60
tctatgaaaa gaactgggaa a 81
<210> 1416
<211> 86
<212> DNA
<213> Brevibacillus agri
<400> 1416
aactgggaaa gtgctaacca cgaagctttc cactaagctt tcgaacgttc aaccttcttc 60
gtgagaaaat tgatgatatt ttgaag 86
<210> 1417
<211> 85
<212> DNA
<213> Brevibacillus agri
<400> 1417
tattttgaag gtgctaacca cgaagctttc cactaagctt tcgaaccgga tcactctcaa 60
cagctccatt acaacgatac ttatt 85
<210> 1418
<211> 83
<212> DNA
<213> Brevibacillus agri
<400> 1418
gatacttatt gtgctaacca cgaagctttc cactaagctt tcgaacgaag ttatgaagaa 60
agtctagcat gtattcccga tgt 83
<210> 1419
<211> 84
<212> DNA
<213> Brevibacillus agri
<400> 1419
ttcccgatgt gtgctaacca cgaagctttc cactaagctt tcgaaccatc aacagcaaca 60
tcaacacgag atcgaggttg aggc 84
<210> 1420
<211> 81
<212> DNA
<213> Brevibacillus agri
<400> 1420
aggttgaggc gtgctaacca cgaagctttc cactaagctt tcgaacctct gtttgcagca 60
gcatacttca aaaagtcttc a 81
<210> 1421
<211> 85
<212> DNA
<213> Brevibacillus agri
<400> 1421
aaagtcttca gtgctaacca cgaagctttc cactaagctt tcgaacataa tccgtttgct 60
gtacggatat aaaaatcttg tatag 85
<210> 1422
<211> 80
<212> DNA
<213> Brevibacillus agri
<400> 1422
tcttgtatag gtgctaacca cgaagctttc cactaagctt tcgaaccgta tgcgaagtct 60
gttatacacc gcattttatg 80
<210> 1423
<211> 57
<212> DNA
<213> Brevibacillus agri
<400> 1423
gcattttatg gtgctaacca cgaagctttc cactaagctt tcgaactcct tcccact 57
<210> 1424
<211> 36
<212> DNA
<213> Brevibacillus agri
<400> 1424
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1425
<211> 87
<212> DNA
<213> Brevibacillus agri
<400> 1425
tggacaagct gtgctaacca cgaagctttc cactaagctt tcgaacttct taacacgcga 60
cgactctttc cccacacgat ttcgatg 87
<210> 1426
<211> 84
<212> DNA
<213> Brevibacillus agri
<400> 1426
gatttcgatg gtgctaatcc cgaagctttc cactaagctt tcgaacaact ataacataac 60
ccaagcaacc gcttatagtt tctg 84
<210> 1427
<211> 86
<212> DNA
<213> Brevibacillus agri
<400> 1427
atagtttctg gtgctaatcc cgaagctttc cactaagctt tcgaacacca gcgccttgtc 60
tgccatgtat tgctgtgcca aagcca 86
<210> 1428
<211> 81
<212> DNA
<213> Brevibacillus agri
<400> 1428
gccaaagcca gtgctaatcc cgaagctttc cactaagctt tcgaactaac cgtaagcctg 60
taacgaagcg ttcgctccaa c 81
<210> 1429
<211> 81
<212> DNA
<213> Brevibacillus agri
<400> 1429
tcgctccaac gtgctaatcc cgaagctttc cactaagctt tcgaactggg acggtgcaca 60
gcagaccgta aaagatgcgt t 81
<210> 1430
<211> 82
<212> DNA
<213> Brevibacillus agri
<400> 1430
aagatgcgtt gtgctaatcc cgaagctttc cactaagctt tcgaacatct agtcacccca 60
aggataattg tcgtcgtagt cc 82
<210> 1431
<211> 84
<212> DNA
<213> Brevibacillus agri
<400> 1431
gtcgtagtcc gtgctaatcc cgaagctttc cactaagctt tcgaacctct agcttgctgt 60
cgaagttagc cctactgttg atga 84
<210> 1432
<211> 84
<212> DNA
<213> Brevibacillus agri
<400> 1432
ctgttgatga gtgctaatcc cgaagctttc cactaagctt tcgaacatct gcatcacggg 60
tagcaacgcc cgcagtcagg tctt 84
<210> 1433
<211> 82
<212> DNA
<213> Brevibacillus agri
<400> 1433
gtcaggtctt gtgctaatcc cgaagctttc cactaagctt tcgaacgagg atcgtgtctg 60
tcaacgggta ttgcacgctt cc 82
<210> 1434
<211> 84
<212> DNA
<213> Brevibacillus agri
<400> 1434
gcacgcttcc gtgctaatcc cgaagctttc cactaagctt tcgaacacgg agtggatttt 60
agagaggggt ggtttttgtt tcta 84
<210> 1435
<211> 81
<212> DNA
<213> Brevibacillus agri
<400> 1435
tttgtttcta gtgctaatcc cgaagctttc cactaagctt tcgaacattt tcgttgccca 60
gacgggagag aggaagagag t 81
<210> 1436
<211> 82
<212> DNA
<213> Brevibacillus agri
<400> 1436
ggaagagagt gtgctaatcc cgaagctttc cactaagctt tcgaactctc ctgatacgcc 60
ccatcagact gcttttctag ca 82
<210> 1437
<211> 83
<212> DNA
<213> Brevibacillus agri
<400> 1437
ttttctagca gtgctaatcc cgaagctttc cactaagctt tcgaactcat atttatcctg 60
taagcttccc tcactcattt tta 83
<210> 1438
<211> 82
<212> DNA
<213> Brevibacillus agri
<400> 1438
ctcattttta gtgctaatcc cgaagctttc cactaagctt tcgaacttga ttgttcggct 60
aaggcgaagg acagagctaa gg 82
<210> 1439
<211> 82
<212> DNA
<213> Brevibacillus agri
<400> 1439
agagctaagg gtgctaatcc cgaagctttc cactaagctt tcgaacgtcc tgacataccg 60
atgttcggcg ctcggtcgaa gt 82
<210> 1440
<211> 86
<212> DNA
<213> Brevibacillus agri
<400> 1440
cggtcgaagt gtgctaatcc cgaagctttc cactaagctt tcgaacgcca ttcgccgagc 60
ctgcggacgt cttcttttcc ggatac 86
<210> 1441
<211> 81
<212> DNA
<213> Brevibacillus agri
<400> 1441
ttccggatac gtgctaatcc cgaagctttc cactaagctt tcgaacccca ggaaacttat 60
atcggcaata gtagctgtct g 81
<210> 1442
<211> 82
<212> DNA
<213> Brevibacillus agri
<400> 1442
tagctgtctg gtgctaatcc cgaagctttc cactaagctt tcgaacgtgc tgcgagtacg 60
agatcaagca ccgcaaggct aa 82
<210> 1443
<211> 85
<212> DNA
<213> Brevibacillus agri
<400> 1443
gcaaggctaa gtgctaatcc cgaagctttc cactaagctt tcgaacttgc cacatttctc 60
tacacggggc tgcgggtcag cgagc 85
<210> 1444
<211> 86
<212> DNA
<213> Brevibacillus agri
<400> 1444
gtcagcgagc gtgctaatcc cgaagctttc cactaagctt tcgaacgtaa cttgcttact 60
caaagtaagc gtgtagtcac ccgccg 86
<210> 1445
<211> 85
<212> DNA
<213> Brevibacillus agri
<400> 1445
tcacccgccg gtgctaatcc cgaagctttc cactaagctt tcgaacggca cgaattcgac 60
ttcgtccgta tgggcgacta tatga 85
<210> 1446
<211> 84
<212> DNA
<213> Brevibacillus agri
<400> 1446
gactatatga gtgctaatcc cgaagctttc cactaagctt tcgaacacta tggcaggatt 60
ggcagaactt tcgccgtttt ttca 84
<210> 1447
<211> 83
<212> DNA
<213> Brevibacillus agri
<400> 1447
cgttttttca gtgctaatcc cgaagctttc cactaagctt tcgaaccccc tttttgacca 60
tgccaagagg aaaagatagc gga 83
<210> 1448
<211> 85
<212> DNA
<213> Brevibacillus agri
<400> 1448
agatagcgga gtgctaatcc cgaagctttc cactaagctt tcgaacttat tttgggtctc 60
gtgagtcgat gcagttgcgt atcta 85
<210> 1449
<211> 82
<212> DNA
<213> Brevibacillus agri
<400> 1449
tgcgtatcta gtgctaatcc cgaagctttc cactaagctt tcgaacccac gtggcgaaag 60
caagttttcc atatcgtcat aa 82
<210> 1450
<211> 81
<212> DNA
<213> Brevibacillus agri
<400> 1450
atcgtcataa gtgctaatcc cgaagctttc cactaagctt tcgaaccttc tattttggga 60
gttggtgctg tgatggtggc g 81
<210> 1451
<211> 82
<212> DNA
<213> Brevibacillus agri
<400> 1451
gatggtggcg gtgctaatcc cgaagctttc cactaagctt tcgaacttct ttggtattca 60
ctccccatcc cgtctcatgg ct 82
<210> 1452
<211> 83
<212> DNA
<213> Brevibacillus agri
<400> 1452
tctcatggct gtgctaatcc cgaagctttc cactaagctt tcgaacgcta gccgtggaca 60
aggaagaaat aaaggaactc att 83
<210> 1453
<211> 82
<212> DNA
<213> Brevibacillus agri
<400> 1453
ggaactcatt gtgctaatcc cgaagctttc cactaagctt tcgaactacc actacctcaa 60
cgctcccgtc gaaaccatca tg 82
<210> 1454
<211> 80
<212> DNA
<213> Brevibacillus agri
<400> 1454
aaccatcatg gtgctaatcc cgaagctttc cactaagctt tcgaactata caggcccgct 60
tttttgatgt cgatgctgga 80
<210> 1455
<211> 82
<212> DNA
<213> Brevibacillus agri
<400> 1455
cgatgctgga gtgctaatcc cgaagctttc cactaagctt tcgaacggtt tcttgatgaa 60
acgcagcgcg tccgtaaagg at 82
<210> 1456
<211> 56
<212> DNA
<213> Brevibacillus agri
<400> 1456
cgtaaaggat gtgctaatcc cgaagctttc cactaagctt tcgaacccct gtcttc 56
<210> 1457
<211> 36
<212> DNA
<213> Brevibacillus agri
<400> 1457
gtgctaatcc cgaagctttc cactaagctt tcgaac 36
<210> 1458
<211> 36
<212> DNA
<213> Brevibacillus agri
<400> 1458
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1459
<211> 397
<212> DNA
<213> Brevibacillus sp.
<400> 1459
gtgctaacca cgaagctttc cactaagctt tcgaacgctt acagcttccg gtcaaccccg 60
catccatccg catagtgcta atcccgaagc tttccactaa gctttcgaac tcaagatcga 120
ggcactgtac ccgggcacga aagggtgcta atcccgaagc tttccactaa gctttcgaac 180
ttaatatctt ggttgtagtt gcgcaaacct tttatgtgct aatcccgaag ctttccacta 240
agctttcgaa ccttactcag tttgcgtcct ccgaagaagt cgtccataag tgctaatccc 300
gaagctttcc actaagcttt cgaacgcgac acgaccgtat tcgtggacga gccggtcttt 360
gtgtgctaat cccgaagctt tccactaagc tttcgaa 397
<210> 1460
<211> 36
<212> DNA
<213> Brevibacillus sp.
<400> 1460
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1461
<211> 80
<212> DNA
<213> Methylobacterium nodulans
<400> 1461
aacagatcgt gtgccaacgc gatcaggatc tggcgcccac tgcgaccccc cgtgtccaca 60
tcgacggccg cgatgcacca 80
<210> 1462
<211> 81
<212> DNA
<213> Methylobacterium nodulans
<400> 1462
cgatgcacca gtgccaacgc gctcaggatc tggcgcccac tgcgacttcc ggcgaccctg 60
gatctgcacg gctacacaac t 81
<210> 1463
<211> 81
<212> DNA
<213> Methylobacterium nodulans
<400> 1463
ctacacaact gtgccaacgc gctcaggatc tggcgcccac tgcgacttgg ccccgccggc 60
accgggtagg agacccgcca t 81
<210> 1464
<211> 79
<212> DNA
<213> Methylobacterium nodulans
<400> 1464
gacccgccat gtgccaacgc gctcaggatc tggcgcccac tgcgacatag ttctggctgt 60
tggaggccag attgttctg 79
<210> 1465
<211> 81
<212> DNA
<213> Methylobacterium nodulans
<400> 1465
gattgttctg gtgccaacgc gctcaggatc tggcgcccac tgcgacctga ccttcacgcg 60
tgtctactcg ggcgtggcca a 81
<210> 1466
<211> 81
<212> DNA
<213> Methylobacterium nodulans
<400> 1466
gcgtggccaa gtgccaacgc gctcaggatc tggcgcccac tgcgacgtcc aggccgagtt 60
gggcaaggtc cgccggatcc t 81
<210> 1467
<211> 56
<212> DNA
<213> Methylobacterium nodulans
<400> 1467
gccggatcct gtgccaacgc gctcaggatc tggcgcccac tgcgacggcg ggcacg 56
<210> 1468
<211> 36
<212> DNA
<213> Methylobacterium nodulans
<400> 1468
gtgccaacgc gctcaggatc tggcgcccac tgcgac 36
<210> 1469
<211> 36
<212> DNA
<213> Methylobacterium nodulans
<400> 1469
gtgccaacgc gctcaggatc tggcgcccac tgcgac 36
<210> 1470
<211> 12
<212> DNA
<213> Methylobacterium nodulans
<400> 1470
gtgccaacgc gc 12
<210> 1471
<211> 24
<212> DNA
<213> Methylobacterium nodulans
<400> 1471
tcaggatctg gcgcccactg cgac 24
<210> 1472
<211> 36
<212> DNA
<213> Alicyclobacillus acidoterrestris
<400> 1472
gtcggatcac tgagcgagcg atctgagaag tggcac 36
<210> 1473
<211> 37
<212> DNA
<213> Desulfonatronum thiodismutans
<400> 1473
gtctcggcaa gcttggtcag tgttgggtga ttggcac 37
<210> 1474
<211> 36
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(36)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1474
ccgcctgacg attcgtgaaa cggcattcgc tgcggc 36
<210> 1475
<211> 36
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1475
gtccaagaaa aaagaaatga tacgaggcat tagcac 36
<210> 1476
<211> 36
<212> DNA
<213> Bacillus sp.
<400> 1476
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1477
<211> 38
<212> DNA
<213> Desulfatirhabdium butyrativorans
<400> 1477
gtgtcagtcg atcaagctgt tttcaccatc ggaacccc 38
<210> 1478
<211> 36
<212> DNA
<213> Brevibacillus agri
<400> 1478
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1479
<211> 36
<212> DNA
<213> Brevibacillus sp.
<400> 1479
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1480
<211> 36
<212> DNA
<213> Methylobacterium nodulans
<400> 1480
gtgccaacgc gctcaggatc tggcgcccac tgcgac 36
<210> 1481
<211> 36
<212> DNA
<213> Alicyclobacillus acidoterrestris
<400> 1481
gtcggatcac tgagcgagcg atctgagaag tggcac 36
<210> 1482
<211> 37
<212> DNA
<213> Desulfonatronum thiodismutans
<400> 1482
gtctcggcaa gcttggtcag tgttgggtga ttggcac 37
<210> 1483
<211> 36
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(36)
<223> /note="Description of Unknown: Opitutaceae bacterium sequence"
<400> 1483
ccgcctgacg attcgtgaaa cggcattcgc tgcggc 36
<210> 1484
<211> 36
<212> DNA
<213> Bacillus thermoamylovorans
<400> 1484
gtccaagaaa aaagaaatga tacgaggcat tagcac 36
<210> 1485
<211> 36
<212> DNA
<213> Bacillus sp.
<400> 1485
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1486
<211> 38
<212> DNA
<213> Desulfatirhabdium butyrativorans
<400> 1486
gtgtcagtcg atcaagctgt tttcaccatc ggaacccc 38
<210> 1487
<211> 36
<212> DNA
<213> Brevibacillus agri
<400> 1487
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1488
<211> 36
<212> DNA
<213> Brevibacillus sp.
<400> 1488
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1489
<211> 36
<212> DNA
<213> Methylobacterium nodulans
<400> 1489
gtcgcagtgg gcgccagatc ctgagcgcgt tggcac 36
<210> 1490
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1490
gtcggatcac tgagcgagcg atctgagaag tggcac 36
<210> 1491
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1491
gtctcggcaa gcttggtcag tgttgggtga ttggcac 37
<210> 1492
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1492
gccgcagcga atgccgtttc acgaatcgtc aggcgg 36
<210> 1493
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1493
gtccaagaaa aaagaaatga tacgaggcat tagcac 36
<210> 1494
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1494
gttcgaaagc ttagtggaaa gcttcgtggt tagcac 36
<210> 1495
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1495
gttcgaaagc ttagtggaaa ttgaagggga tgtacca 37
<210> 1496
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1496
ggggttccga tggtgaaaac agcttgatcg actgac 36
<210> 1497
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1497
gttcgaaagc ttagtggaaa gcttcgtggt tagcac 36
<210> 1498
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1498
gttcgaaagc ttagtggaaa gcttcgtggt tagcac 36
<210> 1499
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1499
gtctcaacgg gcgccagttc ctgagctcgt tggcac 36
<210> 1500
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1500
tgatgtcagc gaaacgacca ccatcggggt tataac 36
<210> 1501
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1501
gggtgcggca tttgcgggtg ttgggggagt ggcagg 36
<210> 1502
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1502
cagtggatgt ttttccatga ggcgaagaat ttcatc 36
<210> 1503
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1503
aacaatataa acgactactt taccgt 26
<210> 1504
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1504
agcttgtcca acttgatgct ccttttcatc 30
<210> 1505
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1505
atcatattca agagcgtgta cactactacg ccgaac 36
<210> 1506
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1506
acagctcggc ctgggtggtg ttggctgcct cggtggcc 38
<210> 1507
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1507
agcttgtcca acttgatgct ccttttcatc 30
<210> 1508
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1508
agcttgtcca acttgatgct ccttttcatc 30
<210> 1509
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1509
tgcatgcgct cctcgaggcg gaggatccga tcgc 34
<210> 1510
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1510
gtcggatcac tgagtgatct aacctatcaa atgcccaaac cacctcctgc ggggttcgaa 60
tcccttcgag tgcgccacaa acataagtaa aacgccggat tcagtttatc gagtccagcg 120
tttttctttt ctactgcagt gttctacgca tcttactatg ccgttgtaat acgaagattt 180
gcgaacatct tggaatacag 200
<210> 1511
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1511
gtctcggcaa gcttggtcag tgatgggtga ttgacatcag tgatgggtga ttgacactag 60
aagcgcacgg cctaggttac gttcttgggg aaagctaggc aagttttgga tgataagaaa 120
taatcatgtc acaaggaggg agtttttgtg gcgagaatca ccatgtatcg aattcaaatc 180
aatacagaag ctataagaaa 200
<210> 1512
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1512
gccgcagtga atgccgtttc accattgatg aagaatgcga ggtgaaaaca gagaaattgg 60
gtcaactcta tcactcttat tcagccatcg tttcaagaaa ggatacctcg tattggatac 120
aacacagctc gttcgttctc tctacctccc tcgacaatct caaggactat ggcgatgcca 180
gcgatggaac agccgaggca 200
<210> 1513
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1513
gttcaagaaa aaagaaatga tatgaggcat tagcacgatg ggatgggaga gagaggacag 60
ttctactctt gctgtatcca gcttctttta ctttatccgg tatcatttct tcacttcttt 120
ctgcacataa aaaagcacct aactatttgg ataagttaag tgcttttatt tccgtttgaa 180
gttgtctatt gcttttttct 200
<210> 1514
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1514
gttcgaaaac ttggtgaaat accttgaaaa ttaacacatc aaagatgccc ctgctttacg 60
ttaggggcat ctttctatta aataacctat agtgaacttc aaaatcattg atctcttgat 120
aataatatat accaaattca atagtcatag tgagaggttt acaggtgctc gatacgtttt 180
cttgcaaaaa agaagcagtc 200
<210> 1515
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1515
gttcgaaagc ttagtggaaa ttgaagggga tgtaccagag gtagcactta gcaagttttt 60
tgagaaacaa gtatctggcc ccaatagcta ggcttatgtg aaatgtagca tgagtggtgt 120
gcaatgaaaa aatagggagg atattagcac tatgaaaaag cagcttaaaa aaacagttaa 180
atgcgtgatg gcgtctatat 200
<210> 1516
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1516
ggggttccga tggtgaaagc agcttgatcg acggacaaat ggatgggcat gcgtaggggc 60
ggttcgcgaa ccgcccctac aactacaccg aaagataccg cgactgaatc tcggtgttgg 120
ccagaagctc ctgcctggtg ccttcgtagc ggatgaggcc tttgtcgatg acatagcccc 180
gatcggagat ggagagggcg 200
<210> 1517
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1517
gttcgaaaac ttggtgaaat accttgaaaa ttaacacatc aaagatgccc ctgctttacg 60
ttaggggcat ctttctatta aataacctat agtgaacttc aaaatcattg atctcttgat 120
aataatatat accaaattca atagtcatag tgagaggttt acaggtgctc gatacgtttt 180
cttgcaaaaa agaagcagtc 200
<210> 1518
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1518
gttcgaaaac ttggtgaaat accttgaaaa ttaacacatc aaagatgccc ctgctttacg 60
ttaggggcat ctttctatta aataacctat agtgaacttc aaaatcattg atctcttgat 120
aataatatat accaaattca atagtcatag tgagaggttt acaggtgctc gatacgtttt 180
cttgcaaaaa agaagcagtc 200
<210> 1519
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1519
gtcgcaacgg atcccagatc ctgatctcgc tggcaggtgc cctggtagat gacgactcgc 60
gctcgttgca gggaggctcg cgcagacgag cgggaaactg cttgtcaggc aatcgcctgt 120
gctgggtccg ctggattctg tgttcgttgg cacgggggag ccgtgggaat gcctactgcc 180
tggatggggt cgcaccaagt 200
<210> 1520
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1520
gttcgaaagc ttagtggaaa ttgaagggga tgtaccc 37
<210> 1521
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1521
gucggaucac ugagcgagcg aucugagaag uggcac 36
<210> 1522
<211> 158
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1522
ucuagaggac agaauuuuuc aacgggugug ccaauggcca cuuuccaggu ggcaaagccc 60
guugagcuuc ucaaaaagaa cgcucgcuca guguucugac cuuucgagcg ccuguucagg 120
gcgaaaaccc ugggaggcgc ucgaaucaua ggugggac 158
<210> 1523
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1523
gccgcagcga augccguuuc acgaaucguc aggcgg 36
<210> 1524
<211> 75
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1524
gcuggagacg uuuuuugaaa cggcgagugc ugcggauagc gaguuucucu uggggaggcg 60
cucgcggcca cuuuu 75
<210> 1525
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1525
guccaagaaa aaagaaauga uacgaggcau uagcac 36
<210> 1526
<211> 107
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1526
cuggacgaug ucucuuuuau uucuuuuuuc uuggaucuga guacgagcac ccacauugga 60
cauuucgcau ggugggugcu cguacuauag guaaaacaaa ccuuuuu 107
<210> 1527
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1527
guucgaaagc uuaguggaaa gcuucguccu uagcac 36
<210> 1528
<211> 69
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1528
cacggauaau cacgacuuuc cacuaagcuu ucgaauuuua ugaugcgagc auccucucag 60
gucaaaaaa 69
<210> 1529
<211> 79
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(79)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1529
gaagaaatga ctattgccaa ctatatctgg cttttctcaa tacccttagc gcgaaaatac 60
cccctcgcca taaccaacc 79
<210> 1530
<211> 84
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(84)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1530
ataaccaacc gtctattgcc atctttatct ggcttttctc aatactatca aggtacagca 60
aatatgctca tcagtgctat gaag 84
<210> 1531
<211> 82
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(82)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1531
tgctatgaag gtctattgcc atctttatct ggcttttctc aatacagtag aaatgattat 60
acctgacaac gttaaagaga gt 82
<210> 1532
<211> 79
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(79)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1532
taaagagagt gtctattgcc atctttatct ggcttttctc aatacaaaag aaaccaaaaa 60
attcattgcg taacaccat 79
<210> 1533
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1533
gtaacaccat gtctattgcc aactatatct ggcttttctc aatacaaaat acgcacagcc 60
tcattaccac tcagtttatt 80
<210> 1534
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1534
tcagtttatt gtctattgcc aactatatct ggcttttctc aatactaaaa tggatgattg 60
gtggctcgat gacactgtcc 80
<210> 1535
<211> 82
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(82)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1535
gacactgtcc gtctattgcc aactatatct ggcttttctc aatacagctt ctagtggtat 60
gccttcggga ccatcaaagg aa 82
<210> 1536
<211> 81
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(81)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1536
atcaaaggaa gtctattgcc aactatatct ggcttttctc aatacgcata tcttgaggat 60
tgggattctg aactcttaga g 81
<210> 1537
<211> 82
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(82)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1537
actcttagag gtctattgcc aactatatct ggcttttctc aatacctcac ttattcacct 60
gggttaactg catgaactca ac 82
<210> 1538
<211> 55
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(55)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1538
tgaactcaac gtctattgcc aactttatct ggcttttctc aataccctac cttct 55
<210> 1539
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1539
gtctattgcc aactatatct ggcttttctc aatac 35
<210> 1540
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1540
gtctattgcc aactatatct ggcttttctc aatac 35
<210> 1541
<211> 78
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(78)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1541
accttccaga gttattgccc tctatcttgg actcttctca tcaactcata ataccaagaa 60
gaaatcaagc atattatc 78
<210> 1542
<211> 80
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(80)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1542
gcatattatc gttattgccc tctatcttgg gctcttctca tcaacacgag ataggcaaac 60
cgaagaacat gatatatata 80
<210> 1543
<211> 78
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(78)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1543
gatatatata gttattgccc tctatcttgg gctcttctca tcaacgaata cgatacaact 60
ggcgactggt ttgattaa 78
<210> 1544
<211> 55
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(55)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1544
gtttgattaa gttattgccc tctatcttgg gctcttctca tcaacaccgt cctct 55
<210> 1545
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1545
gttattgccc tctatcttgg gctcttctca tcaac 35
<210> 1546
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1546
gttattgccc tctatcttgg gctcttctca tcaac 35
<210> 1547
<211> 80
<212> DNA
<213> Clostridium aminophilum
<400> 1547
aaagagcagg gtttggagaa cagcccgata tagagggcaa tagacgcctt ttgtcgcaag 60
tgttgccgta ccggataacg 80
<210> 1548
<211> 80
<212> DNA
<213> Clostridium aminophilum
<400> 1548
ccggataacg gtttggagaa cagcccgata tagagggcaa tagacgcaac atatgcttca 60
tcggggaaga gttctttccc 80
<210> 1549
<211> 80
<212> DNA
<213> Clostridium aminophilum
<400> 1549
gttctttccc gtttggagaa cagcccgata tagagggcaa tagacccgga agcaattccc 60
ccaatggcct gtccaagcca 80
<210> 1550
<211> 79
<212> DNA
<213> Clostridium aminophilum
<400> 1550
gtccaagcca gtttggagaa cagcccgata tagagggcaa tagacttaat tgccggatac 60
ttgagatgga acttgtcac 79
<210> 1551
<211> 78
<212> DNA
<213> Clostridium aminophilum
<400> 1551
aacttgtcac gtttggagaa cagcccgata tagagggcaa tagacatatc atcatcaaga 60
gtctgccttg catcacaa 78
<210> 1552
<211> 79
<212> DNA
<213> Clostridium aminophilum
<400> 1552
tgcatcacaa gtttggagaa cagcccgata tagagggcaa tagacgttta tgtaaactct 60
taacgagtaa ccatcttaa 79
<210> 1553
<211> 81
<212> DNA
<213> Clostridium aminophilum
<400> 1553
accatcttaa gtttggagaa cagcccgata aagagggcaa tagaccagac catacctttt 60
cagacgggat ctttaccatc t 81
<210> 1554
<211> 79
<212> DNA
<213> Clostridium aminophilum
<400> 1554
tttaccatct gtttggagaa cagcccgata aagagggcaa tagacctgat gctgcgaatc 60
tgccagaact cttgcgacc 79
<210> 1555
<211> 56
<212> DNA
<213> Clostridium aminophilum
<400> 1555
tcttgcgacc gtttggagaa cagcccgata tagagggcaa tagacggtgt acttgt 56
<210> 1556
<211> 35
<212> DNA
<213> Clostridium aminophilum
<400> 1556
gtttggagaa cagcccgata tagagggcaa tagac 35
<210> 1557
<211> 35
<212> DNA
<213> Clostridium aminophilum
<400> 1557
gtttggagaa cagcccgata tagagggcaa tagac 35
<210> 1558
<211> 81
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(81)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1558
agaagacagg gttttgagaa tagcccgaca tagagggcaa tagacaacgg ttttaccatt 60
gtcaaactcg cctgctgtcg t 81
<210> 1559
<211> 84
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(84)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1559
ctgctgtcgt gttttgagaa tagcccgaca tagagggcaa tagacttttg cttcgtcacg 60
gatggacttc acaatggcaa caac 84
<210> 1560
<211> 79
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(79)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1560
tggcaacaac gttttgagaa tagcccgaca tagagggcaa tagaccttca taaatccaag 60
atacggatgc atgattacg 79
<210> 1561
<211> 77
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(77)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1561
catgattacg gttttgagaa tagcccgaca tagagggcaa tagactttta aagggatgat 60
aaggaagccg tatactc 77
<210> 1562
<211> 82
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(82)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1562
ccgtatactc gttttgagaa tagcccgaca tagagggcaa tagacctaag ctggtttttc 60
catgttgaca ccttgcctag tt 82
<210> 1563
<211> 81
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(81)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1563
ttgcctagtt gttttgagaa tagcccgaca tagagggcaa tagacttctt ctcttgtcat 60
tcttctacct ctaaaatctc a 81
<210> 1564
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1564
gttttgagaa tagcccgaca tagagggcaa tagac 35
<210> 1565
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1565
gttttgagaa tagcccgaca tagagggcaa tagac 35
<210> 1566
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1566
gttttgagaa tagcccgaca tagagggcaa tagac 35
<210> 1567
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1567
aaaatggaaa gttatagtcc tcttacattt agaggtagtc tttaattaaa agattacaat 60
gaaggttatc cacttc 76
<210> 1568
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1568
tatccacttc gttatagtcc tcttacattt agaggtagtc tttaatttaa ctcatgggat 60
atgaattatc atttag 76
<210> 1569
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1569
tatcatttag gttatagtcc tcttacattt agaggtagtc tttaatgtat taggaacaat 60
aaacgactct ttttta 76
<210> 1570
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1570
ctctttttta gttatagtcc tcttacattt agaggtagtc tttaattcaa attaatcgtt 60
ctctttatat ctggga 76
<210> 1571
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1571
atatctggga gttatagtcc tcttacattt agaggtagtc tttaattgat tatatcgaaa 60
atcaaataaa tgcgct 76
<210> 1572
<211> 37
<212> DNA
<213> Carnobacterium gallinarum
<400> 1572
gttatagtcc tcttacattt agaggtagtc tttaatt 37
<210> 1573
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1573
ccaataaacg gttatagtcc tcttacattt agaggtagtc tttaattctc ctttttcatg 60
aatggccgtt aaccct 76
<210> 1574
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1574
cgttaaccct gttatagtcc tcttacattt agaggtagtc tttaattgcg acaatcagta 60
tgattacgat gctgac 76
<210> 1575
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1575
cgatgctgac gttatagtcc tcttacattt agaggtagtc tttaattttt acataaaaaa 60
aggagggtat aatcat 76
<210> 1576
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1576
gtataatcat gttatagtcc tcttacattt agaggtagtc tttaatttac gccaagaatt 60
attaatgttg atgcaa 76
<210> 1577
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1577
gttgatgcaa gttatagtcc tcttacattt agaggtagtc tttaatcttt atccatgaat 60
taactcatgc gattgc 76
<210> 1578
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1578
atgcgattgc gttatagtcc tcttacattt agaggtagtc tttaatagaa atatattaat 60
agcgacttat attaca 76
<210> 1579
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1579
ttatattaca gttatagtcc tcttacattt agaggtagtc tttaatatat tggagaaaaa 60
agaaaaagat tagtca 76
<210> 1580
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1580
agattagtca gttatagtcc tcttacattt agaggtagtc tttaattcct ctattgtact 60
taacatcatc tactga 76
<210> 1581
<211> 56
<212> DNA
<213> Carnobacterium gallinarum
<400> 1581
catctactga gttatagtcc tcttacattt agaggtagtc tttaataccc ttctat 56
<210> 1582
<211> 36
<212> DNA
<213> Carnobacterium gallinarum
<400> 1582
gttatagtcc tcttacattt agaggtagtc tttaat 36
<210> 1583
<211> 37
<212> DNA
<213> Carnobacterium gallinarum
<400> 1583
gttatagtcc tcttacattt agaggtagtc tttaatt 37
<210> 1584
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1584
tctagttctg attaaagact acccctaaat gtaaggggac tataactcct caatgctttt 60
attttgagcg ttcgct 76
<210> 1585
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1585
agcgttcgct attaaagact acccctaaat gtaaggggac tataactacc gactagagaa 60
ctctttagtc acttga 76
<210> 1586
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1586
agtcacttga attaaagact acccctaaat gtaaggggac tataactgct tttgttaaat 60
tgattattgg gtcttc 76
<210> 1587
<211> 37
<212> DNA
<213> Carnobacterium gallinarum
<400> 1587
attaaagact acccctaaat gtaaggggac tataact 37
<210> 1588
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1588
gttgaggggt aatataaact acctctaaat gtaagaggac tataacttta ttagttgttc 60
atctgttttt ttttaa 76
<210> 1589
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1589
ttttttttaa aatataaact acctctaaat gtaagaggac tataacctac aaaaacgact 60
aaaaaagcgt ttaaag 76
<210> 1590
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1590
gcgtttaaag aatataaact acctctaaat gtaagaggac tataactttg gttgtacccc 60
tttttgaaca tatagg 76
<210> 1591
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1591
aacatatagg aatataaact acctctaaat gtaagaggac tataactgcg taatgatgag 60
gtggcagaat caatgc 76
<210> 1592
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1592
gaatcaatgc aatataaact acctctaaat gtaagaggac tataaccata aaaccttgat 60
tttcataaga tatatc 76
<210> 1593
<211> 76
<212> DNA
<213> Carnobacterium gallinarum
<400> 1593
aagatatatc aatataaact acctctaaat gtaagaggac tataacccaa tagtcaattc 60
tgttagccag taggga 76
<210> 1594
<211> 57
<212> DNA
<213> Carnobacterium gallinarum
<400> 1594
ccagtaggga aatataaact acctctaaat gtaagaggac tataactgct atataaa 57
<210> 1595
<211> 36
<212> DNA
<213> Carnobacterium gallinarum
<400> 1595
aatataaact acctctaaat gtaagaggac tataac 36
<210> 1596
<211> 37
<212> DNA
<213> Carnobacterium gallinarum
<400> 1596
attaaagact acccctaaat gtaaggggac tataact 37
<210> 1597
<211> 75
<212> DNA
<213> Paludibacter propionicigenes
<400> 1597
gcatgtcctt gttttagttc ccttcaattt tgggataatc cacaagtatt actgctgcaa 60
tgcagattgg cgtat 75
<210> 1598
<211> 75
<212> DNA
<213> Paludibacter propionicigenes
<400> 1598
attggcgtat gttgtagttc ccttcaattt tgggataatc cacaagtatt actgctgcaa 60
tgcagattgg cgtat 75
<210> 1599
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1599
attggcgtat gttgtagttc ccttcaattt tgggataatc cacaaggaaa acgaaaatta 60
taacttcaat aaattc 76
<210> 1600
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1600
caataaattc gttgtagttc cctttaattt tgggataatc cacaagagta aaaccgtaca 60
atggagaaga taacgt 76
<210> 1601
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1601
aagataacgt gatgtagttc ccttcaattt tgggatagtc cacaagagcg gtgcggattt 60
gagcgatgcg aatttg 76
<210> 1602
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1602
tgcgaatttg gttgtagttc ccttcaatat tgggataatc cacaaggaat taggattatg 60
agtttatata gtttga 76
<210> 1603
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1603
tatagtttga gttgtagttc ccttcaatat tgggataatt cacaagtaaa cattaaaaca 60
ataaacggaa gttcga 76
<210> 1604
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1604
ggaagttcga gttgtagttc ccttcaattt tgggataatc cataagaaag caaaaatgaa 60
cacaaaacaa tttgaa 76
<210> 1605
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1605
acaatttgaa gttttagttc ccttcgattt tgggataatc cacaagtaca gcagtaagcg 60
gaaacactca ggcaat 76
<210> 1606
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1606
ctcaggcaat gttgtagttc ccttcaattt tgggataatc cacaaggccg taaaaaatcc 60
acacaacgat aagatg 76
<210> 1607
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1607
cgataagatg gttttagttc ccttcaattt tgggataatc cataagctct tactggctgg 60
tgccgggctt gctgca 76
<210> 1608
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1608
gcttgctgca gttgtagttc ccttcaattt tgggattatc cacaagagaa agagtgatga 60
acgacgagca ggatat 76
<210> 1609
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1609
agcaggatat gttgtagttc ccttcaattt tgggattatc cacaagttac acctgatcaa 60
tggaaattgc ttagtg 76
<210> 1610
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1610
ttgcttagtg gttgtagttc ccttcaattt tgggataatc cacaagtgtg attaatgata 60
ttaccgtagc taacgg 76
<210> 1611
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1611
tagctaacgg gttgtagttc ccttcattat tgggatagtc cacaagaaaa caacctgcat 60
ggataaatta tatgac 76
<210> 1612
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1612
attatatgac gttctagctc ccttcaattt tgggataatc cacaagtaaa aacatatttg 60
ctgaagacta tggaag 76
<210> 1613
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1613
actatggaag gttgtagttc ccttcaatat tgggataatc cacaagtgct attcggaata 60
gatttttacc ccacca 76
<210> 1614
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1614
taccccacca gttgtagttc ccttcaattt tgggataatt cacaagtggg attatgaaat 60
aaccgattat aaacta 76
<210> 1615
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1615
ttataaacta gttgtagttc ccttcaattt tgggataatt cacaagagaa atcaaaaatt 60
gattcttcag tagctt 76
<210> 1616
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1616
tcagtagctt gttgtagttc ccttcaattt tgggataatt cacaagcgaa tcctaatggt 60
tatattactg cttcat 76
<210> 1617
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1617
actgcttcat gttgtagttc ccttcaattt tgggataatt cacaagtaaa ttgaaaattt 60
ctggacaagc tccagt 76
<210> 1618
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1618
aagctccagt gttgtagttc ccttcaatat tgggataatc cacaagaaac gaatcacaga 60
acatgtgaac ctggta 76
<210> 1619
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1619
gaacctggta gttgtagttc ccttcaattt tgggataatc cacaagagat gataacgtag 60
gcaacatccc tgataa 76
<210> 1620
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1620
tccctgataa gttttagttc ccttcaattt tgggataatc cacaagcgtt attcaggaga 60
gagtttcaaa gagtat 76
<210> 1621
<211> 76
<212> DNA
<213> Paludibacter propionicigenes
<400> 1621
caaagagtat gttttagttc ccttcaatat tgggataatc cacaagtaaa acattggcta 60
ataacagaac tagggc 76
<210> 1622
<211> 56
<212> DNA
<213> Paludibacter propionicigenes
<400> 1622
gaactagggc gttttagttc ccttcaatat tgggataatc cacaagatag tatctg 56
<210> 1623
<211> 36
<212> DNA
<213> Paludibacter propionicigenes
<400> 1623
gttgtagttc ccttcaattt tgggataatc cacaag 36
<210> 1624
<211> 36
<212> DNA
<213> Paludibacter propionicigenes
<400> 1624
gttgtagttc ccttcaattt tgggataatc cacaag 36
<210> 1625
<211> 36
<212> DNA
<213> Paludibacter propionicigenes
<400> 1625
gttgtagttc ccttcaattt tgggataatc cacaag 36
<210> 1626
<211> 36
<212> DNA
<213> Paludibacter propionicigenes
<400> 1626
gttgtagttc ccttcaattt tgggataatc cacaag 36
<210> 1627
<211> 76
<212> DNA
<213> Listeria seeligeri
<400> 1627
tgttattgca gtaagagact acctctatat gaaagaggac taaaaccata tttccaaact 60
ccactttgac tacacc 76
<210> 1628
<211> 76
<212> DNA
<213> Listeria seeligeri
<400> 1628
tgactacacc gtaagagact acctctatat gaaagaggac taaaacggtc ccactacttg 60
aggtacgaac atatca 76
<210> 1629
<211> 76
<212> DNA
<213> Listeria seeligeri
<400> 1629
gaacatatca gtaagagact acctctatat gaaagaggac taaaacttag tcaacccctc 60
gctgcatttt cacatt 76
<210> 1630
<211> 76
<212> DNA
<213> Listeria seeligeri
<400> 1630
ttttcacatt gtaagagact acctctatat gaaagaggac taaaacgatg gataataggg 60
atagatcatt agtccg 76
<210> 1631
<211> 56
<212> DNA
<213> Listeria seeligeri
<400> 1631
cattagtccg gtaagagact acctctatat gaaagaggac taaaacgtct aatgtg 56
<210> 1632
<211> 36
<212> DNA
<213> Listeria seeligeri
<400> 1632
gtaagagact acctctatat gaaagaggac taaaac 36
<210> 1633
<211> 36
<212> DNA
<213> Listeria seeligeri
<400> 1633
gtaagagact acctctatat gaaagaggac taaaac 36
<210> 1634
<211> 76
<212> DNA
<213> Listeria weihenstephanensis
<400> 1634
tattttcata gatttagagt acctcaaaac agaagaggac taaaacgcac tctccgacaa 60
taatctcgtc catttt 76
<210> 1635
<211> 76
<212> DNA
<213> Listeria weihenstephanensis
<400> 1635
cgtccatttt gatttagagt acctcaaaac aaaagaggac taaaacaact ctgtacttgt 60
gaagtacgtt aaatcc 76
<210> 1636
<211> 56
<212> DNA
<213> Listeria weihenstephanensis
<400> 1636
cgttaaatcc gatttagagt acctcaaaac aaaagaggac taaaacctct tttgtg 56
<210> 1637
<211> 36
<212> DNA
<213> Listeria weihenstephanensis
<400> 1637
gatttagagt acctcaaaac aaaagaggac taaaac 36
<210> 1638
<211> 76
<212> DNA
<213> Listeria weihenstephanensis
<400> 1638
gtctacaagt gatttagagt agctcaaaaa agaagaggtc taaaacagag atactgatac 60
tattgagccc agtcat 76
<210> 1639
<211> 76
<212> DNA
<213> Listeria weihenstephanensis
<400> 1639
gcccagtcat gatttagagt acctcaaaat agaagaggtc taaaactgtt ccaaggcttg 60
ctaactcacc tttttc 76
<210> 1640
<211> 76
<212> DNA
<213> Listeria weihenstephanensis
<400> 1640
cacctttttc gatttagagt acctcaaaat agaagaggtc taaaacatct tgtttatatc 60
cgtatcggca cccaaa 76
<210> 1641
<211> 76
<212> DNA
<213> Listeria weihenstephanensis
<400> 1641
ggcacccaaa gatttagagt gcctcaaaat agaagaggtc taaaactcac tctccatcca 60
ctcaatcaga tcattt 76
<210> 1642
<211> 76
<212> DNA
<213> Listeria weihenstephanensis
<400> 1642
cagatcattt gatttagagt acctcaaaat agaagaggtc taaaactatc aatttccttt 60
tgatttctta aatcag 76
<210> 1643
<211> 76
<212> DNA
<213> Listeria weihenstephanensis
<400> 1643
cttaaatcag gatttagagt acctcaaaat agaagaggtc taaaacgttc caatatggtt 60
ttttccacat cttccg 76
<210> 1644
<211> 57
<212> DNA
<213> Listeria weihenstephanensis
<400> 1644
acatcttccg gatttagagt acctcaaaat agaagaggtc taaaacctca aattgaa 57
<210> 1645
<211> 36
<212> DNA
<213> Listeria weihenstephanensis
<400> 1645
gatttagagt acctcaaaat agaagaggtc taaaac 36
<210> 1646
<211> 36
<212> DNA
<213> Listeria weihenstephanensis
<400> 1646
gatttagagt acctcaaaac aaaagaggac taaaac 36
<210> 1647
<211> 36
<212> DNA
<213> Listeria weihenstephanensis
<400> 1647
gatttagagt acctcaaaac aaaagaggac taaaac 36
<210> 1648
<211> 75
<212> DNA
<213> Listeria newyorkensis
<400> 1648
cgtaatgctt gatttagagt acctcaaaac aaaagaggac taaaactact tgtcgatatg 60
gtatagcttt tttca 75
<210> 1649
<211> 76
<212> DNA
<213> Listeria newyorkensis
<400> 1649
gcttttttca gatttagagt acctcaaaac aaaagaggac taaaactaaa gcttctaaat 60
ggtggcgcgt tacgcc 76
<210> 1650
<211> 76
<212> DNA
<213> Listeria newyorkensis
<400> 1650
gcgttacgcc gatttagaat acctcaaaac aaaagaggac taaaacctag caagccggtc 60
gccgcgctca aagtaa 76
<210> 1651
<211> 57
<212> DNA
<213> Listeria newyorkensis
<400> 1651
ctcaaagtaa gatttagagt acctcaaaac aaaagaggac taaaacctct tttgtgg 57
<210> 1652
<211> 36
<212> DNA
<213> Listeria newyorkensis
<400> 1652
gatttagagt acctcaaaac aaaagaggac taaaac 36
<210> 1653
<211> 36
<212> DNA
<213> Listeria newyorkensis
<400> 1653
gatttagagt acctcaaaac aaaagaggac taaaac 36
<210> 1654
<211> 36
<212> DNA
<213> Listeria newyorkensis
<400> 1654
gatttagagt acctcaaaac aaaagaggac taaaac 36
<210> 1655
<211> 78
<212> DNA
<213> Leptotrichia wadei
<400> 1655
atgatgtatg taagttttag tccccttcgt ttttggggta gtctaaatcc tggtaaacca 60
atccacatcg aagaaaag 78
<210> 1656
<211> 76
<212> DNA
<213> Leptotrichia wadei
<400> 1656
cgaagaaaag aaagttttag tccccttcgt ttttggggta gtctaaatca tgtagaagaa 60
gttattgtat ctattt 76
<210> 1657
<211> 59
<212> DNA
<213> Leptotrichia wadei
<400> 1657
gtatctattt tacgttttag tccccttcgt ttttggggta gtctaaatct ttatatcaa 59
<210> 1658
<211> 39
<212> DNA
<213> Leptotrichia wadei
<400> 1658
taagttttag tccccttcgt ttttggggta gtctaaatc 39
<210> 1659
<211> 39
<212> DNA
<213> Leptotrichia wadei
<400> 1659
taagttttag tccccttcgt ttttggggta gtctaaatc 39
<210> 1660
<211> 39
<212> DNA
<213> Leptotrichia wadei
<400> 1660
taagttttag tccccttcgt ttttggggta gtctaaatc 39
<210> 1661
<211> 78
<212> DNA
<213> Leptotrichia shahii
<400> 1661
attctttaga gttttagtcc ccttcgatat tggggtggtc tatatcgaaa aagaagagtt 60
tattcagata gatttgtc 78
<210> 1662
<211> 77
<212> DNA
<213> Leptotrichia shahii
<400> 1662
tagatttgtc gttttagtcc ccttcgatat tggggtggtc tatatcaata tggattactt 60
ggtagaacag caatcta 77
<210> 1663
<211> 57
<212> DNA
<213> Leptotrichia shahii
<400> 1663
cagcaatcta gttttagtcc ccttcgatat tggggtggtc tatatcccat cctaatt 57
<210> 1664
<211> 36
<212> DNA
<213> Leptotrichia shahii
<400> 1664
gttttagtcc ccttcgatat tggggtggtc tatatc 36
<210> 1665
<211> 36
<212> DNA
<213> Leptotrichia shahii
<400> 1665
gttttagtcc ccttcgatat tggggtggtc tatatc 36
<210> 1666
<211> 80
<212> DNA
<213> Rhodobacter capsulatus
<400> 1666
gaacatcatg ggttcagtcc gccgtcgtct tggcggtgat gtgaggctct cccagcatac 60
caaaccgctg gcgaccatca 80
<210> 1667
<211> 78
<212> DNA
<213> Rhodobacter capsulatus
<400> 1667
gcgaccatca ggttcagtcc gccgtcgtct tggcggtgat gtgatgcaca ggagagaccg 60
atgaaaatca cggccttc 78
<210> 1668
<211> 79
<212> DNA
<213> Rhodobacter capsulatus
<400> 1668
cacggccttc ggttcagtcc gccgtcgtct tggcggtgac gtgaggcacc taaacaagag 60
gttctacgat gccgaaagg 79
<210> 1669
<211> 58
<212> DNA
<213> Rhodobacter capsulatus
<400> 1669
tgccgaaagg ggttcagtcc gccgtcgtct tggcggtgat gtgaggctca ggtcccgc 58
<210> 1670
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1670
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1671
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1671
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1672
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1672
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1673
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1673
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1674
<211> 80
<212> DNA
<213> Rhodobacter capsulatus
<400> 1674
gaacatcatg ggttcagtcc gccgtcgtct tggcggtgat gtgaggctct cccagcatac 60
caaaccgctg gcgaccatca 80
<210> 1675
<211> 78
<212> DNA
<213> Rhodobacter capsulatus
<400> 1675
gcgaccatca ggttcagtcc gccgtcgtct tggcggtgat gtgatgcaca ggagagaccg 60
atgaaaatca acggcttc 78
<210> 1676
<211> 79
<212> DNA
<213> Rhodobacter capsulatus
<400> 1676
caacggcttc ggttcagtcc gccgtcgtct tggcggtgac gtgaggcacc taaacaagag 60
gttctacgat gccgaaagg 79
<210> 1677
<211> 58
<212> DNA
<213> Rhodobacter capsulatus
<400> 1677
tgccgaaagg ggttcagtcc gccgtcgtct tggcggtgat gtgaggctca ggtcccgc 58
<210> 1678
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1678
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1679
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1679
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1680
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1680
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1681
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1681
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1682
<211> 80
<212> DNA
<213> Rhodobacter capsulatus
<400> 1682
gaacatcatg ggttcagtcc gccgtcgtct tggcggtgat gtgaggctct cccagcatac 60
caaaccgctg gcgaccatca 80
<210> 1683
<211> 78
<212> DNA
<213> Rhodobacter capsulatus
<400> 1683
gcgaccatca ggttcagtcc gccgtcgtct tggcggtgat gtgatgcaca ggagagaccg 60
atgaaaatca acggcttc 78
<210> 1684
<211> 79
<212> DNA
<213> Rhodobacter capsulatus
<400> 1684
caacggcttc ggttcagtcc gccgtcgtct tggcggtgac gtgaggcacc taaacaagag 60
gttctacgat gccgaaagg 79
<210> 1685
<211> 58
<212> DNA
<213> Rhodobacter capsulatus
<400> 1685
tgccgaaagg ggttcagtcc gccgtcgtct tggcggtgat gtgaggctca ggtcccgc 58
<210> 1686
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1686
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1687
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1687
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1688
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1688
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1689
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1689
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1690
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1690
gtctattgcc aactatatct ggcttttctc aatac 35
<210> 1691
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1691
gttattgccc tctatcttgg gctcttctca tcaac 35
<210> 1692
<211> 35
<212> DNA
<213> Clostridium aminophilum
<400> 1692
gtttggagaa cagcccgata tagagggcaa tagac 35
<210> 1693
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1693
gttttgagaa tagcccgaca tagagggcaa tagac 35
<210> 1694
<211> 37
<212> DNA
<213> Carnobacterium gallinarum
<400> 1694
gttatagtcc tcttacattt agaggtagtc tttaatt 37
<210> 1695
<211> 36
<212> DNA
<213> Carnobacterium gallinarum
<400> 1695
aatataaact acctctaaat gtaagaggac tataac 36
<210> 1696
<211> 36
<212> DNA
<213> Paludibacter propionicigenes
<400> 1696
gttgtagttc ccttcaattt tgggataatc cacaag 36
<210> 1697
<211> 36
<212> DNA
<213> Listeria seeligeri
<400> 1697
gtaagagact acctctatat gaaagaggac taaaac 36
<210> 1698
<211> 36
<212> DNA
<213> Listeria weihenstephanensis
<400> 1698
gatttagagt acctcaaaac aaaagaggac taaaac 36
<210> 1699
<211> 36
<212> DNA
<213> Listeria newyorkensis
<400> 1699
gatttagagt acctcaaaac aaaagaggac taaaac 36
<210> 1700
<211> 39
<212> DNA
<213> Leptotrichia wadei
<400> 1700
taagttttag tccccttcgt ttttggggta gtctaaatc 39
<210> 1701
<211> 36
<212> DNA
<213> Leptotrichia shahii
<400> 1701
gttttagtcc ccttcgatat tggggtggtc tatatc 36
<210> 1702
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1702
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1703
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1703
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1704
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1704
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1705
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1705
gtctattgcc aactatatct ggcttttctc aatac 35
<210> 1706
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1706
gttattgccc tctatcttgg gctcttctca tcaac 35
<210> 1707
<211> 35
<212> DNA
<213> Clostridium aminophilum
<400> 1707
gtttggagaa cagcccgata tagagggcaa tagac 35
<210> 1708
<211> 35
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(35)
<223> /note="Description of Unknown: Lachnospiraceae bacterium
sequence"
<400> 1708
gttttgagaa tagcccgaca tagagggcaa tagac 35
<210> 1709
<211> 37
<212> DNA
<213> Carnobacterium gallinarum
<400> 1709
gttatagtcc tcttacattt agaggtagtc tttaatt 37
<210> 1710
<211> 36
<212> DNA
<213> Carnobacterium gallinarum
<400> 1710
aatataaact acctctaaat gtaagaggac tataac 36
<210> 1711
<211> 36
<212> DNA
<213> Paludibacter propionicigenes
<400> 1711
gttgtagttc ccttcaattt tgggataatc cacaag 36
<210> 1712
<211> 36
<212> DNA
<213> Listeria seeligeri
<400> 1712
gtaagagact acctctatat gaaagaggac taaaac 36
<210> 1713
<211> 36
<212> DNA
<213> Listeria weihenstephanensis
<400> 1713
gatttagagt acctcaaaac aaaagaggac taaaac 36
<210> 1714
<211> 36
<212> DNA
<213> Listeria newyorkensis
<400> 1714
gatttagagt acctcaaaac aaaagaggac taaaac 36
<210> 1715
<211> 39
<212> DNA
<213> Leptotrichia wadei
<400> 1715
taagttttag tccccttcgt ttttggggta gtctaaatc 39
<210> 1716
<211> 36
<212> DNA
<213> Leptotrichia shahii
<400> 1716
gttttagtcc ccttcgatat tggggtggtc tatatc 36
<210> 1717
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1717
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1718
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1718
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1719
<211> 37
<212> DNA
<213> Rhodobacter capsulatus
<400> 1719
ggttcagtcc gccgtcgtct tggcggtgat gtgaggc 37
<210> 1720
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1720
gtattgagaa aagccagata tagttggcaa tagtc 35
<210> 1721
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1721
attaaagact acctctaaat gtaagaggac tataac 36
<210> 1722
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1722
gttatagtcc tcttacattt agaggtagtc tttaat 36
<210> 1723
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1723
attaaagact acctctaaat gtaagaggac tataac 36
<210> 1724
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1724
gttttagtcc tcttctgttt tgaggtactc taaatc 36
<210> 1725
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1725
gttttagccc tcttttgttc tgaggtactc taaatc 36
<210> 1726
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1726
gttttgagaa tagcccgaca tagagggcaa tagac 35
<210> 1727
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1727
gtttggagaa cagcccgata tagagggcaa tagac 35
<210> 1728
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1728
tcacatcacc gccaagacga cggcggactg aacc 34
<210> 1729
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1729
tcacatcacc gccaagacga cggcggactg aacc 34
<210> 1730
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1730
tcacatcacc gccaagacga cggcggactg aacc 34
<210> 1731
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1731
gctggagaag atagcccaag aaagagggca ataac 35
<210> 1732
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1732
gttttagtcc tcttttgttt tgaggtactc taaatc 36
<210> 1733
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1733
gatatagacc accccaatat cgaaggggac taaaac 36
<210> 1734
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1734
tggattatcc caaaattgaa gggaacta 28
<210> 1735
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1735
gttttagtcc tctttcatat agaggtagtc tcttac 36
<210> 1736
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1736
atttcttctc ttgtcattct tctacctttc gcacaa 36
<210> 1737
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1737
tttccatttt cccactactt tctaaaaaga 30
<210> 1738
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1738
tgattatatc gaaaatcaaa taaatgcgct 30
<210> 1739
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1739
cgtttattgg tcagagtaaa ctcaactccg 30
<210> 1740
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1740
tatgaaaata atgtaacacc aatcgtttgg 30
<210> 1741
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1741
atattttttg taacggcttg caatcattt 29
<210> 1742
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1742
ttcttctctt gtcattcttc tacctctaaa atctca 36
<210> 1743
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1743
ggtgtacttg ttccactcaa tccacttatc atctt 35
<210> 1744
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1744
catgatgttc ctttcttggg tatggggtaa gcc 33
<210> 1745
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1745
catgatgttc ctttcttggg tatggggtaa gcc 33
<210> 1746
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1746
catgatgttc ctttcttggg tatggggtaa gcc 33
<210> 1747
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1747
gctaacatct ccggtgttat taccactcca ttct 34
<210> 1748
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1748
aagcattacg gcgtatcacg ccaccaatta 30
<210> 1749
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1749
tctaaagaat tatctattct gtcttttaaa tt 32
<210> 1750
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1750
aaacaaggac atgcacatac ccacatgttt ttctcttg 38
<210> 1751
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1751
tgcaataaca ttttctgata cttttggcgg 30
<210> 1752
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1752
gtattgagaa aagccagata tagcatgaac acttcggtgt ttgtgctttt ttagtatgac 60
gggcatgccg tcagtctgtg gtgaaagtcc acaaggggcg tagttgccaa cgaaccccaa 120
agcaactccc aaggtttaca ccgtgaggtg taggcgagaa gaagggatag caaaatcgta 180
gcctgacgaa cagaaacctg 200
<210> 1753
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1753
attaaagact acctctaaat gtaagaaaaa taaaaaaata aaagagttac atatagttaa 60
gaaaaagaag taggaatatt tattcctact tctttttcgt tgtatttaat ttatttatat 120
gaaaatatga taagatagat atatagtatt agaatggagg gtattgttaa gatgcgtata 180
acaaaagtga aaataaaatt 200
<210> 1754
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1754
gttatagtcc tcttacattt agaggttgga atggcaacag tttttttgac aaattttata 60
aggtgcagaa cttctttccg tatgctattc cgattgtcct tgacaatgag cctcctcgga 120
tgtgacgatc ttcggcagga gctaacagtt aaagttagac tgcctcgcta gcgttagcac 180
atccgagctc attatcaagg 200
<210> 1755
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1755
attaaagact acctggatag gctacaatta actagacaga aaaattaagg tgtgtagact 60
agaagaaaac ataccaggag gaatttttat gtctaagaga acacgaagaa ctttttcaca 120
agaattcaag caacaaatcg tcaatcttta cttagctgga aagccacgtg tagaaatcat 180
tcgagaatat gaactaacgg 200
<210> 1756
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1756
gttttagtcc tcttctgttt tgaggtaata tatccgccta ttcgcttgca acaacataac 60
tataccagat tattaatgaa tctgcagctt gacagattag caaaacataa ttattttttc 120
ataaattaag agacagctca gcagccatct cttaaattca accaattatc ttctgctaac 180
aaaatacctt ccttcttgaa 200
<210> 1757
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1757
gttttagtcc tcttctgttt tgaggtattt tttaatagca aaatgaaatt gcattctccc 60
atccaatttc attttgaaat taactgcaac attctatatc aaatttctaa tagtctcttt 120
actataccac aatactcttc tgaaacttga tttgtttcta tataaccatt atcgattttt 180
ttctcaccta gatgtctcaa 200
<210> 1758
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1758
gttttgagaa tagcccgaca tagttataga gatgtataaa tataaccgat aaacattgac 60
taatttgttg aagtcagtgt ttatcggttt tttgtgtaaa tataggagtt gttagaatga 120
tactttttgc ctaattttgg aactttatga ggatataaga tagacttgat aaaaaggtaa 180
aagaaaggtt aaagagcatg 200
<210> 1759
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1759
gtttgtagaa cagcctgata tagagggcga taggactttg gctgcatgac tcgatcatta 60
agcctgaaac taagttttct tgtgttgaaa tcttcctaat actgaggtcg taagaccatc 120
ttgattattc acgaatctgt gactctgttc tgagaacaat ctcatactat aaggacaatg 180
ttttttgaaa tggaggattt 200
<210> 1760
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1760
tcacatcacc gccaagatga cggcgggaac ccaatgcaaa cggaggtgcg gcaacagcaa 60
ggttgcaccg gctggacttc ggcggcagtc tggcgcaacg gggcgcagga cacggaagat 120
gtggcggggg caagatggac ctgtttttca aggccacgga atatgagacc ctgcaggcct 180
catggctcaa ggtccagcaa 200
<210> 1761
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1761
tcacatcacc gccaagatga cggcgggaac ccaatgcaaa cggaggtgcg gcaacagcaa 60
ggttgcaccg gctggacttc ggcggcagtc tggcgcaacg gggcgcagga cacggaagat 120
gtggcggggg caagatggac ctgtttttca aggccacgga atatgagacc ctgcaggcct 180
catggctcaa ggtccagcaa 200
<210> 1762
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1762
tcacatcacc gccaagatga cggcgggaac ccaatgcaaa cggaggtgcg gcaacagcaa 60
ggttgcaccg gctggacttc ggcggcagtc tggcgcaacg gggcgcagga cacggaagat 120
gtggcggggg caagatggac ctgtttttca aggccacgga atatgagacc ctgcaggcct 180
catggctcaa ggtccagcaa 200
<210> 1763
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1763
gttggagaag agagcccaag atagaggaga ttgacattta ttacaagcgg agattaaaac 60
gataactgag aaaaaatgaa taacgctgat gaaaacggcc ggattcttgg ccgttttttt 120
gtctatttgc taagtgcaca aagattgtga aataacatct gctactatgt atttatcgag 180
gtacgtaaat ctaggtggtg 200
<210> 1764
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1764
gttttagtcc tcttttattt tgaggtaata tattcacttg caacaacata actataccag 60
attattaatg gatctgcatc ttgacagatt accaaaacat aattattttt tcataaatta 120
agagacaact cagaatacag aattgcctct acatgtctaa ttttcccact gtcaattcct 180
ctgcgcataa tatctccacc 200
<210> 1765
<211> 200
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1765
gatatagacc accccaatat cgaaaagtga tatttaataa aaataacttc tgagtgagaa 60
taaaatttca attcttgctc attttttatt gttttttgaa tatggttgct tggttgttct 120
gaaacgaaaa tttttggaga tgttttaaat ttttaggttg aaaaaaaata agaattatac 180
tataataaat aattatgcga 200
<210> 1766
<211> 250
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1766
tggattatcc caaaattgaa gggtaacact acagctgaca tcaaagcaca aacaaccccg 60
aatgaaattc atcattcggg gttgttttta taaaggttag cttagctaat tgcagtccta 120
cagcaaatca ctacttcttc aaacgcaata tctccggatt ttctgcaata aatttattgg 180
cagcttcgta gccctgatga tagatagtga agtaagagtc gtagctaaac tcatgaaagg 240
tttcgatggc 250
<210> 1767
<211> 250
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1767
gttttagtcc tctttcattt agaggtatat cgtattccta cttaataata gtaattaaaa 60
caaccaatgt aaaggatata atcaatatat ttaaagtttg cacgagaatg caatcatttt 120
attcataaat atcatatcat ttataagctc tattttccat tttctaaggc taataaataa 180
aactgctgta cctatggatc taaggaagac ttatgcacac agtacagcaa cttttcagca 240
tgatttgtgt 250
<210> 1768
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1768
auuauuacca uuuugguugg aaugcuauua uaaaggauca uucgauuauu accucuaccu 60
cccuucccac gauuucuu 78
<210> 1769
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1769
gcuggagaag auagcccaag aaagagggca auaac 35
<210> 1770
<211> 81
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1770
gucuuacgac cucaguauua ggaagauuuc aaccaagaaa acuuaguuuc aggcuuaaug 60
aucgagucau gcagccaaag u 81
<210> 1771
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1771
guuuggagaa cagcccgaua uagagggcaa uagac 35
<210> 1772
<211> 99
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1772
augaaaagag gacuaaaacu gaaagaggac uaaaacacca gauguggaua acuauauuag 60
uggcuauuaa aaauucgucg auauuagaga ggaaacuuu 99
<210> 1773
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1773
guuuuagucc ucuuucauau agagguaguc ucuuac 36
<210> 1774
<211> 106
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1774
uuaguauacc acaucaauau uaaaucucaa aaaaauaagg agccgucaaa cauagcuccc 60
uacuucuauu uacucauaau ccccaucuau ccuuacuuuu cguaaa 106
<210> 1775
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1775
guuuuagucc ccuucgauau uggggugguc uauauc 36
<210> 1776
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1776
cccataattg ataggatcta tgaggt 26
<210> 1777
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1777
ctcccgaaaa gccttgt 17
<210> 1778
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1778
cccatgattg ataggatcta tgaggt 26
<210> 1779
<211> 18
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1779
tttcccccga caggcgta 18
<210> 1780
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1780
cccataattg ataggatcta tgaggt 26
<210> 1781
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1781
tccatatgaa tggcgcg 17
<210> 1782
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1782
cccataattg ataggatcta tgaggt 26
<210> 1783
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1783
tgccgccgtc ctgcatg 17
<210> 1784
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1784
cccataattg ataggatcta tgaggt 26
<210> 1785
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1785
gcccggacca catgcac 17
<210> 1786
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1786
cccataattg ataggatcta tgaggt 26
<210> 1787
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1787
aaatataaat acattaa 17
<210> 1788
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1788
cccataattg ataggatcta tgaggt 26
<210> 1789
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1789
atcggttata caggcta 17
<210> 1790
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1790
cccataattg ataggatcta tgagga 26
<210> 1791
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1791
acggcgccgg aaaacat 17
<210> 1792
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1792
cccataattg ataggatcta tgaggc 26
<210> 1793
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1793
agagccgcgc ctattgg 17
<210> 1794
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1794
cccataattg ataggatcta tgaggt 26
<210> 1795
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1795
aaaacggtcc cactaat 17
<210> 1796
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1796
cccataattg ataggatcta tgaggt 26
<210> 1797
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1797
aaaacggtcc cactaat 17
<210> 1798
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1798
cccataattg ataggatcta tgaggt 26
<210> 1799
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1799
tgagctgctg gcccgca 17
<210> 1800
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1800
cccataattg ataggatcta tgagga 26
<210> 1801
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1801
tggttccaat gcagtaa 17
<210> 1802
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1802
cccataattg ataggatcta tgaggt 26
<210> 1803
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1803
acctataacg gcaccta 17
<210> 1804
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1804
cccataattg ataggatcta tgaggc 26
<210> 1805
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1805
cccataattg ataggatcta tgaggt 26
<210> 1806
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1806
cccataattg ataggatcta tgaggt 26
<210> 1807
<211> 18
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(18)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1807
ttgccgccgt cctgcatg 18
<210> 1808
<211> 27
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(27)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1808
cacgtagttt tgaagggaag ttagagg 27
<210> 1809
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1809
accgaatatg agaactg 17
<210> 1810
<211> 27
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(27)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1810
atcgtagttt tgaagggaag ttagagg 27
<210> 1811
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1811
tacaaataaa catcaag 17
<210> 1812
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1812
tcgtagtttt gaagggaagt tagagg 26
<210> 1813
<211> 16
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1813
ccgctccggc cgtgat 16
<210> 1814
<211> 27
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(27)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1814
atcgtagttt tgaagggaag ttagagg 27
<210> 1815
<211> 27
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(27)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1815
atcgtagttt tgaagggaag ttagagg 27
<210> 1816
<211> 27
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(27)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1816
atcgtagttt tgaagggaag ttagagg 27
<210> 1817
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1817
gttagaattg agaggatgtt gaagga 26
<210> 1818
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1818
ttacacggcc tatggtc 17
<210> 1819
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1819
gttagaattg agaggatgtt gaaggt 26
<210> 1820
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1820
cttatgcaca acccttt 17
<210> 1821
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1821
gttagaattg agaggatgtt gaagga 26
<210> 1822
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1822
aactgaacgg cttgttt 17
<210> 1823
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1823
gttagaattg agaggatgtt gaagga 26
<210> 1824
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1824
tcttactaat ttgccga 17
<210> 1825
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1825
gttagaattg agaggatgtt gaagga 26
<210> 1826
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1826
gttagaattg agaggatgtt gaagga 26
<210> 1827
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1827
gttagaattg agaggatgtt gaagga 26
<210> 1828
<211> 17
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(17)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1828
aactgaacgg cttgttt 17
<210> 1829
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1829
cccataattg ataggatcta tgaggt 26
<210> 1830
<211> 27
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(27)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1830
atcgtagttt tgaagggaag ttagagg 27
<210> 1831
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1831
gttagaattg agaggatgtt gaagga 26
<210> 1832
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Gut metagenome sequence"
<400> 1832
cccataattg ataggatcta tgaggt 26
<210> 1833
<211> 27
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(27)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1833
atcgtagttt tgaagggaag ttagagg 27
<210> 1834
<211> 26
<212> DNA
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(26)
<223> /note="Description of Unknown: Marine metagenome sequence"
<400> 1834
gttagaattg agaggatgtt gaagga 26
<210> 1835
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1835
cctctaactt cccttcaaaa ctacgat 27
<210> 1836
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1836
cagttctcat attcggt 17
<210> 1837
<211> 225
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1837
cctctaactt cccttcaaaa ctacgtgaca aaggttaaca tattgtaatt ccacttgatt 60
ttgtcatacc agggagaaaa ttggcatgga aatggatcaa tttcagcacc ccgattcggt 120
ttggacatgg aaatctaacc aacggggccg caaggcaagt ctgtggattc cctacctcga 180
tcaaataaga aaaattaagg ggactcgctg ggaatttgtt tataa 225
<210> 1838
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1838
gtctcggcaa gcttggtcag tgttgggtga ttggcac 37
<210> 1839
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1839
ccgcctgacg attcgtgaaa cggcattcgc tgcggc 36
<210> 1840
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1840
cttcaacgaa gccacgccgc gaacggcgtg gagtg 35
<210> 1841
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1841
gcttcaacga agccacgccg cgaacggcgt ggagtg 36
<210> 1842
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1842
gtccaagaaa aaagaaatga tacgaggcat tagcac 36
<210> 1843
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1843
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1844
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1844
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1845
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1845
gtgctaacca cgaagctttc cactaagctt tcgaac 36
<210> 1846
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1846
gtgtcagtcg atcaagctgt tttcaccatc ggaacccc 38
<210> 1847
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1847
gtgctaatcc cgaagctttc cactaagctt tcgaac 36
<210> 1848
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1848
gtgccaacgc gctcaggatc tggcgcccac tgcgac 36
<210> 1849
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1849
gtttcaatcc gcgcccccgt gagggggc 28
<210> 1850
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1850
cggatcatcc ccgcatccgc gggggacac 29
<210> 1851
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1851
gtttcaatcc gcgcccccgt gagagggcga c 31
<210> 1852
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1852
gtctattgcc aactatatct ggcttttctc aatac 35
<210> 1853
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1853
gtctattgcc atctttatct ggcttttctc aatac 35
<210> 1854
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1854
gttattgccc tctatcttgg gctcttctca tcaac 35
<210> 1855
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1855
gttattgccc tctttcttgg gctatcttct ccagc 35
<210> 1856
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1856
gtttggagaa cagcccgata tagagggcaa tagac 35
<210> 1857
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1857
gttttgagaa tagcccgaca tagagggcaa tagac 35
<210> 1858
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1858
gttatagtcc tcttacattt agaggtagtc tttaat 36
<210> 1859
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1859
gttatagtcc tcttacattt agaggtagtc tttaat 36
<210> 1860
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1860
gttcttaatc taaaatagtg aaatgtaaat 30
<210> 1861
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1861
gcatttacat tacattattt tagattaaga ac 32
<210> 1862
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1862
actacattat agctgattct gtaaggaaac tatagc 36
<210> 1863
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1863
actacattat agctgattct gtaaggaaac tatagc 36
<210> 1864
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1864
tagttccctt caattttggg attatcca 28
<210> 1865
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1865
cctttcaaac gaagggcact tacaac 26
<210> 1866
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1866
gttttaacta cttattgtga aatgtaaat 29
<210> 1867
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1867
gttttaatta cttattgtga aatgtaaat 29
<210> 1868
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1868
gtaagagact acctctatat gaaagaggac taaaac 36
<210> 1869
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1869
gatttagagt acctcaaaac agaagaggac taaaac 36
<210> 1870
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1870
gatttagagt agctcaaaaa agaagaggtc taaaac 36
<210> 1871
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1871
gatttagagt acctcaaaac aaaagaggac taaaac 36
<210> 1872
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1872
gttttagtcc ccttcgtttt tggggtagt 29
<210> 1873
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1873
tcaatcctta ttttaatgga ttcactattc ttac 34
<210> 1874
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1874
gttttaatag cacaaattgt attgtaaat 29
<210> 1875
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1875
gtttcaatcc ttgttttaat ggataactta ctttaac 37
<210> 1876
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1876
gtttcaatcc ttgttttaat ggatactcta ctttaac 37
<210> 1877
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1877
gttttagtcc ccttcgatat tggggtggtc tatatc 36
<210> 1878
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1878
acctctcccg ccagtaagcg gattgagac 29
<210> 1879
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1879
gcccagacac ctctcccgcc agtaagcgga ttgagac 37
<210> 1880
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1880
gcccagacac ctctcccgcc agtaagcgga ttgagac 37
<210> 1881
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1881
gtcgctcccc ccgcgggggc gtggatcgaa ac 32
<210> 1882
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1882
gtcgctcccc ccgcgggggc gtggatcgaa ac 32
<210> 1883
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1883
ggttcagtcc gccgtcgtct tggcggtgat gtga 34
<210> 1884
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1884
gccctctcct cctccctggg ccg 23
<210> 1885
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1885
ggttcagtcc gccgtcgtct tggcggtgat gtga 34
<210> 1886
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1886
ggttcagtcc gccgtcgtct tggcggtgat gtga 34
<210> 1887
<211> 3994
<212> DNA
<213> Listeria weihenstephanensis
<400> 1887
tggctgctga gctgtctctt aatttatgaa aaaataatta tgttttgcta atctgtcaag 60
ctgcagattc attaataatc tggtatagtt atgttgttgc aagcgaatag gcggatatat 120
tacctcaaaa cagaagagga ctaaaaccca aacgattggt gttacattat tttcatagat 180
ttagagtacc tcaaaacaga agaggactaa aacgcactct ccgacaataa tctcgtccat 240
tttgatttag agtacctcaa aacaaaagag gactaaaaca actctgtact tgtgaagtac 300
gttaaatccg atttagagta cctcaaaaca aaagaggact aaaacctctt ttgtggataa 360
gtattcgaaa taaagccata aaaactgtga tccaaagaac tggattattg gtttttatgg 420
ctttattcaa ttcttagtat tgtagatgaa ctgtcagcga atgttgtctt gcaacgtgcc 480
ttcttgtata atgaatatat tgataaataa tagaaatttc atacacgcat gaagaaacca 540
attaaagttt cagcaataat gaagcattag gtacatgact ataaaaccaa atggagctga 600
gtagacagat gaaaatcaca aagatgagag tagatggaag aactatcgta atggagagga 660
caagtaagga aggtcaactg gtttatgaag gtatcgatgg aaataagaca acagaaatta 720
tatttgataa gaaaaaagaa tcgttttata agagtatcct caataaaact gtgagaaaac 780
ctgatgaaaa aagaaaaaaa ataggcgtaa gcaggcaatt aataaagcga ttaataaaga 840
aataacagaa ttaatgttgg cgctgttaca tcaagaagtg ccaagccaaa agttacataa 900
tttaaagagt ctaaatacgg aatctttaac taaactattt aaaccgaagt tccaaaacat 960
gatttcttat ccgcctagca aaggtgccga acatgttcaa ttttgcctta cagatatagc 1020
ggtaccagcg attcgagatt tagatgaaat taagccagat tggggcattt tttttgaaaa 1080
attgaaaccc tatacggatt gggcagaatc atacattcac tataagcaga caaccataca 1140
gaaatccatt gagcaaaaca aaatacagtc ccctgattcg ccaaggaaat tagtattgca 1200
aaaatatgtc acagcctttt tgaatggaga accgctggga ctcgatcttg tggcgaaaaa 1260
atataaactg gcagacttag cggagtcgtt taaagtagta gatttgaacg aggataaaag 1320
tgcaaactat aaaattaaag cgtgcttgca acaacatcag cgaaatattt tggatgaatt 1380
gaaagaagat ccagagttaa atcaatatgg tatagaagtg aagaagtata tacagcgata 1440
tttcccaatc aaacgtgcac cgaatagaag taaacatgcg cgagcggact ttttgaaaaa 1500
ggaattaatt gagtctacag tggagcagca atttaaaaat gctgtatatc attatgtact 1560
ggaacaagga aaaatggagg catatgagct aacagatcct aaaacaaaag acttgcagga 1620
tattagatct ggtgaggcat ttagcttcaa atttattaat gcttgcgcct tcgcatccaa 1680
taatttgaag atgattttaa accctgaatg tgaaaaggat attttaggta agggcgattt 1740
taaaaagaat ttgccaaaca gtactacgca gtctgatgtt gtgaaaaaaa tgattccttt 1800
tttctcggat gagattcaaa atgtgaattt tgatgaagct atctgggcga ttaggggctc 1860
tattcagcaa attagaaatg aggtttacca ttgcaaaaag cattcttgga aaagcatact 1920
taaaataaaa ggctttgaat ttgaacctaa caatatgaaa tatacggatt ctgatatgca 1980
aaaattgatg gataaagata tcgccaaaat tccagacttc atcgaagaaa aacttaaaag 2040
tagtgggata ataaggttct acagtcatga taaattgcag tctatctggg aaatgaagca 2100
agggttttcg ttgttgacta ctaatgcgcc gtttgtccca agctttaaac gtgtctacgc 2160
aaaagggcac gactaccaaa cttctaaaaa tagatattat gatttaggtt tgactacttt 2220
tgatattttg gaatatggag aagaagattt tcgtgcacgc tatttcctga cgaagctagt 2280
ttattatcaa caatttatgc catggtttac agctgataat aatgctttcc gagatgctgc 2340
caattttgta ttgcgattaa ataaaaatag acagcaggat gcaaaagctt ttattaacat 2400
tagagaagtt gaagaaggtg agatgcctag agactatatg ggctatgtcc aaggtcaaat 2460
agcgatacat gaggattcaa ctgaggatac accgaatcat tttgaaaaat ttattagcca 2520
ggtttttatt aagggatttg atagtcatat gagatctgct gatttaaaat ttattaaaaa 2580
tccaagaaat caggggctag aacaaagtga aattgaggaa atgagctttg atattaaagt 2640
agagccatca tttttgaaaa ataaagatga ctatattgca ttttggacat tctgcaaaat 2700
gctggatgct aggcatttaa gcgagctaag aaacgaaatg attaagtatg acggtcattt 2760
aactggagaa caagaaatca ttggtttagc attgcttgga gtggattcac gagagaatga 2820
ttggaagcaa ttttttagct cagaacggga atacgagaaa attatgaagg gctatgttgg 2880
agaggaattg tatcagcggg aaccgtaccg acaaagtgat ggcaaaacac cgattctttt 2940
tcgtggtgta gagcaagcga ggaagtatgg tactgaaaca gtgattcaac ggctttttga 3000
tgctagtcct gagtttaaag tgtcgaaatg caacataact gagtgggagc ggcaaaaaga 3060
aaccattgaa gagactattg agcgaagaaa agaattgcat aatgaatggg aaaaaaatcc 3120
caaaaaaccg caaaataatg cattttttaa agagtataaa gagtgttgtg acgctattga 3180
tgcttacaat tggcataaaa ataaaactac gcttgtatac gttaatgagc tgcaccattt 3240
gctaattgaa attctgggaa gatatgttgg ctatgtagca atagctgata gagactttca 3300
atgtatggcg aatcaatatt ttaagcattc aggaataact gagagagtgg aatattgggg 3360
cgataataga ctaaaaagta ttaaaaagct ggatacattc ttgaaaaaag aaggactgtt 3420
tgtttctgag aaaaatgcaa ggaatcatat agcgcattta aattatttat cactcaaatc 3480
tgagtgcacg ttgctgtatt tatctgagag gttgagagaa atttttaagt atgatcgtaa 3540
attaaagaat gccgtttcca agtcattaat cgatatttta gatagacatg gtatgagcgt 3600
cgtatttgct aacttgaaag aaaataaaca taggttggtg ataaaaagct tagagccaaa 3660
aaaattgaga catctaggtg agaaaaaaat cgataatggt tatatagaaa caaatcaagt 3720
ttcagaagag tattgtggta tagtaaagag actattagaa atttgatata gaatgttgca 3780
gttaatttca aaatgaaatt ggatgggaga atgcaatttc attttgctat taaaaaatac 3840
ctcaaaacag aagaggacta aaacaaatga ttgcaagccg ttacaaaaaa tatgatttag 3900
agtacctcag aacaaaagag ggctaaaact agaataatcg agtgagacgg tctacaagtg 3960
atttagagta gctcaaaaaa gaagaggtct aaaa 3994
<210> 1888
<211> 4053
<212> DNA
<213> Listeria newyorkensis
<400> 1888
tgtattctga gttgtctctt aatttatgaa aaaataatta tgttttggta atctgtcaag 60
atgcagatcc attaataatc tggtatagtt atgttgttgc aagtgaatat attacctcaa 120
aataaaagag gactaaaact aattggtggc gtgatacgcc gtaatgcttg atttagagta 180
cctcaaaaca aaagaggact aaaactactt gtcgatatgg tatagctttt ttcagattta 240
gagtacctca aaacaaaaga ggactaaaac taaagcttct aaatggtggc gcgttacgcc 300
gatttagaat acctcaaaac aaaagaggac taaaacctag caagccggtc gccgcgctca 360
aagtaagatt tagagtacct caaaacaaaa gaggactaaa acctcttttg tggataagta 420
ttcgaaataa agccataaaa actgtgatcc aaagagctgg attattggtt tttatggctt 480
tattcaattc ttagtattgt agatgaacca tcagtgaatg ttgtcttgca acgtgccttc 540
ttgtataatg aatatattga taaataatag aaatttcata cacgcatgaa gaaaccaatt 600
aaattttcag cattaatgaa gcattaggta catgactata aaaccaaatg gagctgagta 660
gacagatgaa aatcacaaag atgagagtag atggaagaac tatcgtaatg gagaggacaa 720
gcaaggaagg tcaactgggt tatgaaggta tcgatggaaa taagacaaca gaaattatat 780
ttgataagaa aaaagagtca ttttataaga gtatcctcaa taaaactgtg agaaaacccg 840
atgaaaaaga aaagaatagg cgtaagcagg caattaataa agcgattaat aaagaaataa 900
cagaattaat gttggcggtg ttacatcaag aagtgccaag ccaaaagtta cataatttaa 960
agagtctaaa tacggaatct ttaactaaac tatttaaacc gaagttccaa aacatgattt 1020
cttatccgcc tagcaaaggt gccgaacatg ttcagttttg ccttacagat atagcggtac 1080
cagcgattcg agatttagat gaaattaagc cagattgggg catttttttt gaaaaattga 1140
aaccctatac ggattgggca gaatcataca ttcactataa gcagacaacc atacagaaat 1200
ccattgagca aaacaaaata cagtcccctg attcgccaag gaaattagta ttgcaaaaat 1260
atgtcacagc ctttttgaat ggagaaccgc tgggactcga tcttgtggcg aaaaaatata 1320
aactggcaga cttagcggag tcgtttaaat tagtagattt gaacgaggat aaaagtgcaa 1380
actataaaat taaagcgtgc ttgcaacaac atcagcgaaa tattttggat gaattgaaag 1440
aagatccgga gttaaatcaa tatggtatag aagtgaagaa atatatacag cgatatttcc 1500
caatcaaacg tgcaccgaat agaagtaaac atgcacgagc ggactttttg aaaaaggaat 1560
taattgagtc tacagtggag caacaattta aaaatgctgt atatcattat gtactggaac 1620
aaggcaaaat ggaggcatat gagctaacag atcctaaaac aaaagacttg caggatatta 1680
gatctggtga ggcatttagc ttcaaattta ttaatgcttg cgccttcgca tcgaataatt 1740
tgaagatgat tttaaaccct gaatgtgaaa aggatatcct aggtaagggc aattttaaaa 1800
agaatttgcc aaacagtact acgcgatctg atgttgtgaa gaaaatgatt ccctttttct 1860
cggatgagct tcaaaatgtg aattttgatg aagctatctg ggcgattagg ggctctattc 1920
agcaaattag aaatgaggtt taccattgta aaaagcattc ttggaaaagc atacttaaaa 1980
taaaaggctt tgaatttgaa cctaacaata tgaaatatgc ggattctgat atgcaaaaat 2040
tgatggataa agatatcgcc aaaattccag agttcatcga agaaaaactt aaaagtagtg 2100
gagtagtaag gttctacagg catgatgagt tgcaatccat atgggaaatg aaacaaggat 2160
tttcgttgct gactactaat gcgccgtttg tcccaagctt taagcgtgtc tacgcaaaag 2220
ggcacgacta ccaaacttct aaaaatagat actataattt ggatttgact acttttgata 2280
ttttggaata tggagaagaa gattttcgtg cacgctattt cctgacgaag ctagtttatt 2340
atcagcaatt tatgccatgg tttacagctg ataataatgc tttccgagat gctgccaatt 2400
ttgtattgcg attaaataaa aatagacagc aggatgcaaa agcttttatt aacattagag 2460
aagttgaaga aggtgagatg cctagagact atatgggtta tgtccaaggt caaatagcga 2520
tacatgagga ttcaattgag gatacaccga atcattttga gaaatttatt agtcaggttt 2580
ttattaaggg ctttgatagg catatgagat ctgctaattt aaaatttatt aaaaatccaa 2640
gaaatcaggg gctagaacaa agtgagattg aggaaatgag ctttgatatt aaagtggagc 2700
cgtcattttt gaaaaataaa gatgactata ttgcattttg gatattctgc aaaatgcttg 2760
atgctaggca tttaagcgag ctaagaaacg aaatgattaa gtatgacggt catttaactg 2820
gagaacaaga aatcattggt ttagcattgc tcggagtgga ttcacgagag aatgattgga 2880
agcagttttt tagctcagaa cgggaatacg agaaaattat gaagggctat gttgtagagg 2940
aattgtatca gcgggaaccg taccgacaaa gtgatggcaa aacaccgatt ctttttcgtg 3000
gtgtagagca agcgaggaag tatggtactg aaacagtgat tcaacggctt tttgatgcta 3060
atcctgagtt caaagtgtca aaatgcaact tagcagagtg ggagcggcaa aaagaaacca 3120
ttgaagagac tattaagcga agaaaagaat tgcataatga atgggcaaaa aatccaaaaa 3180
aaccgcaaaa taatgcattt tttaaagagt ataaagagtg ttgtgacgct attgacgctt 3240
acaattggca taaaaataaa actacgcttg catacgttaa tgagctgcac catttgctaa 3300
ttgaaattct gggaagatat gttggctatg tagcaatagc tgatagagac tttcaatgta 3360
tggcgaatca atattttaag cattcaggaa taactgagag agtggaatat tggggcgata 3420
atagactaaa aagtattaaa aagctggata cattcttgaa aaaagaagga ctgtttgttt 3480
ctgagaaaaa tgcaaggaat catatagcgc atttaaatta tttatcactc aaatctgagt 3540
gcacgttgct gtatttatcc gagaggttga gagaaatttt taagtatgat cgtaaattaa 3600
agaatgccgt ttccaagtca ttaatcgata ttttagatag acatggtatg agcgtcgtat 3660
ttgctaactt gaaagaaaat aaacataggt tggtgataaa aagcttagag ccaaaaaaat 3720
tgagacatct gggtgggaaa aaaatcgatg gtggttatat agaaacaaat caagtttcag 3780
aagagtattg tggtatagtg aagagactgt tagaaatgtg atgtaggatg ttacagtcaa 3840
tctcaaaatg aaattggaag ttacaagagg acgtaatatg atgttactat taaaaatacc 3900
tcagaataga agaggactaa aacaaatgat tgcaagccgt tacaaaaaat atgatttaga 3960
gtacctcaga acaaaagagg gctaaaacta gaataatcga ttgagacggt ctacaagtga 4020
tttagagtac ctcaaaacag aagaggacta aaa 4053
<210> 1889
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1889
attaaagact acctctaaat gtaagaggac tataac 36
<210> 1890
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1890
attaaagact acctctaaat gtaagaggac tataac 36
<210> 1891
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1891
attaaagact acccctaaat gtaaggggac tataac 36
<210> 1892
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1892
aatataaact acctctaaat gtaagaggac tataac 36
<210> 1893
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1893
acuaccucua uaugaaagag gacuaaaac 29
<210> 1894
<211> 47
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> misc_feature
<222> (1)..(47)
<223> /note="Variant nucleotides given in the sequence have no
preference with respect to those in the annotations for variant
positions"
<220>
<221> modified_base
<222> (30)..(47)
<223> a, c, u, g, unknown or other
<220>
<221> variation
<222> (45)..(47)
<223> /replace=" "
<400> 1894
acuaccucua uaugaaagag gacuaaaacn nnnnnnnnnn nnnnnnn 47
<210> 1895
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(56)
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(28)
<223> a, c, u, g, unknown or other
<220>
<221> misc_feature
<222> (1)..(56)
<223> /note="Variant nucleotides given in the sequence have no
preference with respect to those in the annotations for variant
positions"
<220>
<221> variation
<222> (15)..(28)
<223> /replace=" "
<400> 1895
nnnnnnnnnn nnnnnnnnnn nnnnnnnncc accccaauau cgaaggggac uaaaac 56
<210> 1896
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(56)
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> modified_base
<222> (1)..(28)
<223> a, c, u, g, unknown or other
<220>
<221> misc_feature
<222> (1)..(56)
<223> /note="Variant nucleotides given in the sequence have no
preference with respect to those in the annotations for variant
positions"
<220>
<221> variation
<222> (15)..(28)
<223> /replace=" "
<400> 1896
nnnnnnnnnn nnnnnnnnnn nnnnnnnncc accccaauau cgaaggggac uaaaac 56
<210> 1897
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1897
ccaccccaau aucgaagggg acuaaaac 28
<210> 1898
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(56)
<223> /note="Description of Artificial Sequence: Synthetic
oligonucleotide"
<220>
<221> misc_feature
<222> (1)..(56)
<223> /note="Variant nucleotides given in the sequence have no
preference with respect to those in the annotations for variant
positions"
<220>
<221> modified_base
<222> (29)..(56)
<223> a, c, u, g, unknown or other
<220>
<221> variation
<222> (43)..(56)
<223> /replace=" "
<400> 1898
ccaccccaau aucgaagggg acuaaaacnn nnnnnnnnnn nnnnnnnnnn nnnnnn 56
<210> 1899
<211> 50
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1899
aggugaaggu cguccguacg aagguaccca gaccgcuaaa cugaaaguua 50
<210> 1900
<211> 65
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1900
atatcaatat ggattacttg gtagaacagc aatctagttt tagtcccctt cgatattggg 60
gtgct 65
<210> 1901
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1901
ccgucugucg uauccagcug caaacuucc 29
<210> 1902
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1902
gucguaucca gcugcaaacu uccagacaa 29
<210> 1903
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1903
guuagauggc cgucugucgu auccagcug 29
<210> 1904
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1904
cagacaacgu gcaacauauc gcgacguau 29
<210> 1905
<211> 70
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1905
uacccgggga uccucuagaa auauggauua cuugguagaa cagcaaucua cucgaccugc 60
aggcaugcaa 70
<210> 1906
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1906
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 1907
<211> 58
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1907
ggccagugaa uucgagcucg guacccgggg auccucuaga aauauggauu acuuggua 58
<210> 1908
<211> 79
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1908
gaacagcaau cuacucgacc ugcaggcaug caagcuuggc guaaucaugg ucauagcugu 60
uuccuguguu uauccgcuc 79
<210> 1909
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1909
acaauuccac acaacauacg agccggaagc auaaag 36
<210> 1910
<211> 53
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1910
gugaauucga gcucgguacc cggggauccu cuagaaauau ggauuacuug gua 53
<210> 1911
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1911
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 1912
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1912
uagauugcug uucuaccaag uaau 24
<210> 1913
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1913
uagauugcug uucuaccaag uaa 23
<210> 1914
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1914
uagauugcug uu 12
<210> 1915
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1915
ccaccccaau aucgaagggg acuaaaac 28
<210> 1916
<211> 36
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1916
gauauagacc accccaauau cgaaggggac uaaaac 36
<210> 1917
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1917
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 1918
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1918
ccacccgaau aucgaacggg acuaaaac 28
<210> 1919
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1919
ccaccgcaau aucgaagcgg acuaaaac 28
<210> 1920
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1920
ccacgccaau aucgaaggcg acuaaaac 28
<210> 1921
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1921
ccagcccaau aucgaagggc acuaaaac 28
<210> 1922
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1922
ccaggggaau aucgaacccc acuaaaac 28
<210> 1923
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1923
ccaccaccaa uaucgaaggg gacuaaaac 29
<210> 1924
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1924
ccaacccaau aucgaagggg acuaaaac 28
<210> 1925
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1925
ccacccaaau aucgaagggg acuaaaac 28
<210> 1926
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1926
ccacccccaa uaucgaaggg ggacuaaaac 30
<210> 1927
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1927
ccaccccaau aucgaagggg acuaaaac 28
<210> 1928
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1928
ccacccgaau aucgaacggg acuaaaac 28
<210> 1929
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1929
ccaccgcaau aucgaagcgg acuaaaac 28
<210> 1930
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1930
ccacgccaau aucgaaggcg acuaaaac 28
<210> 1931
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1931
ccagcccaau aucgaagggc acuaaaac 28
<210> 1932
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1932
ccaggggaau aucgaacccc acuaaaac 28
<210> 1933
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1933
ccaccaccaa uaucgaaggg gacuaaaac 29
<210> 1934
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1934
ccaacccaau aucgaagggg acuaaaac 28
<210> 1935
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1935
ccacccaaau aucgaagggg acuaaaac 28
<210> 1936
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1936
ccacccccaa uaucgaaggg ggacuaaaac 30
<210> 1937
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1937
ccaccccaau aucgaagggg acuaaaac 28
<210> 1938
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1938
ccaccccaua ucgaagggga cuaaaac 27
<210> 1939
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1939
ccaccccauc gaaggggacu aaaac 25
<210> 1940
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1940
ccaccccaag gggacuaaaa c 21
<210> 1941
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1941
ccaccccaaa uaucgaaggg gacuaaaac 29
<210> 1942
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1942
ccaccccaaa aauaucgaag gggacuaaaa c 31
<210> 1943
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1943
ccaccccaaa aaaaaauaug gaaggggacu aaaac 35
<210> 1944
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1944
ccaccccgau aucgaagggg acuaaaac 28
<210> 1945
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1945
ccaccccaaa aucgaagggg acuaaaac 28
<210> 1946
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1946
ccaccccaau auccaagggg acuaaaac 28
<210> 1947
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1947
ccaccccaau aucgaagggg acuaaaac 28
<210> 1948
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1948
ccaccccaua cugaagggga cuaaaac 27
<210> 1949
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1949
ccaccccaug caaggggacu aaaac 25
<210> 1950
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1950
ccaccccaag gggacuaaaa c 21
<210> 1951
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1951
ccaccccaaa uuacgaaggg gacuaaaac 29
<210> 1952
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1952
ccaccccaaa aaauucgaag gggacuaaaa c 31
<210> 1953
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1953
ccaccccaaa aaaaaauauc gaaggggacu aaaac 35
<210> 1954
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1954
ccaccccgau aucgaagggg acuaaaac 28
<210> 1955
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1955
ccaccccaaa aucgaagggg acuaaaac 28
<210> 1956
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1956
ccaccccaau auccaagggg acuaaaac 28
<210> 1957
<211> 55
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1957
aucaaguuag auggccgucu gucguaucca gcugcaaacu uccagacaac gugca 55
<210> 1958
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1958
ccaccccaau aucgaagggg acuaaaacaa guuugcagcu ggauacgaca gacggc 56
<210> 1959
<211> 33
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1959
ggtccgctgc cgttcgcttg ggacatcctg tcc 33
<210> 1960
<211> 70
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1960
ataggccagt gaattcgagc tcgaatatgg attacttggt agaacagcaa tctacgccgg 60
aagcataaag 70
<210> 1961
<211> 173
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 1961
ggccagtgaa ttcgagctcg gtacccgggg atcctctaga aatatggatt acttggtaga 60
acaccaatct actcgacctg caggcatgca agcttggcgt aatcatggtc atagctgttt 120
cctgtgttta tccgctcaca attccacaca acatacgagc cggaagcata aag 173
<210> 1962
<211> 16
<212> PRT
<213> Lactococcus lactis
<400> 1962
Leu Gln Lys Phe Thr Gly Asp Ile Glu Asn Leu Val Lys Ala Ser Leu
1 5 10 15
<210> 1963
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 1963
Trp Ile Arg Ala Gly Trp Phe Ile Arg Asn Arg Ser Ala His Tyr
1 5 10 15
<210> 1964
<211> 16
<212> PRT
<213> Lactococcus lactis
<400> 1964
Leu Gln Lys Phe Thr Gly Asp Ile Glu Asn Leu Val Lys Ala Ser Leu
1 5 10 15
<210> 1965
<211> 15
<212> PRT
<213> Lactococcus lactis
<400> 1965
Trp Ile Arg Ala Gly Trp Phe Ile Arg Asn Arg Ser Ala His Tyr
1 5 10 15
<210> 1966
<211> 16
<212> PRT
<213> Thermus aquaticus
<400> 1966
Pro Leu Glu Glu Arg Gly Val Ala Glu Ala Trp Leu Asn Ala Lys Val
1 5 10 15
<210> 1967
<211> 10
<212> PRT
<213> Thermus aquaticus
<400> 1967
Ala Asp Leu Arg Asn Asp Val Ala His Ala
1 5 10
<210> 1968
<211> 16
<212> PRT
<213> Thermus aquaticus
<400> 1968
Pro Leu Glu Glu Arg Gly Val Ala Glu Ala Trp Leu Asn Ala Lys Val
1 5 10 15
<210> 1969
<211> 10
<212> PRT
<213> Thermus aquaticus
<400> 1969
Ala Asp Leu Arg Asn Asp Val Ala His Ala
1 5 10
<210> 1970
<211> 16
<212> PRT
<213> Mycobacterium tuberculosis
<400> 1970
Arg Ser Ala Thr Pro Ala Ile Thr Ile Val Leu Arg Ala Ala Val Ala
1 5 10 15
<210> 1971
<211> 15
<212> PRT
<213> Mycobacterium tuberculosis
<400> 1971
Leu Gly Arg Phe Glu Ser Arg Val Arg Asn Thr Ala Ala His Glu
1 5 10 15
<210> 1972
<211> 16
<212> PRT
<213> Mycobacterium tuberculosis
<400> 1972
Arg Ser Ala Thr Pro Ala Ile Thr Ile Val Leu Arg Ala Ala Val Ala
1 5 10 15
<210> 1973
<211> 15
<212> PRT
<213> Mycobacterium tuberculosis
<400> 1973
Leu Gly Arg Phe Glu Ser Arg Val Arg Asn Thr Ala Ala His Glu
1 5 10 15
<210> 1974
<211> 16
<212> PRT
<213> Escherichia coli
<400> 1974
Leu Tyr Pro Glu Leu Arg Thr Ile Glu Gly Val Leu Lys Ser Lys Met
1 5 10 15
<210> 1975
<211> 14
<212> PRT
<213> Escherichia coli
<400> 1975
Ala Tyr Thr Phe Phe Asn Val Glu Arg His Ser Leu Phe His
1 5 10
<210> 1976
<211> 16
<212> PRT
<213> Escherichia coli
<400> 1976
Leu Tyr Pro Glu Leu Arg Thr Ile Glu Gly Val Leu Lys Ser Lys Met
1 5 10 15
<210> 1977
<211> 14
<212> PRT
<213> Escherichia coli
<400> 1977
Ala Tyr Thr Phe Phe Asn Val Glu Arg His Ser Leu Phe His
1 5 10
<210> 1978
<211> 16
<212> PRT
<213> Homo sapiens
<400> 1978
Gly Asn Glu Leu Pro Leu Ala Val Ala Ser Thr Ala Asp Leu Ile Arg
1 5 10 15
<210> 1979
<211> 15
<212> PRT
<213> Homo sapiens
<400> 1979
Ile Pro Asp Trp Ile Val Asp Leu Arg His Glu Leu Thr His Lys
1 5 10 15
<210> 1980
<211> 16
<212> PRT
<213> Homo sapiens
<400> 1980
Gly Asn Glu Leu Pro Leu Ala Val Ala Ser Thr Ala Asp Leu Ile Arg
1 5 10 15
<210> 1981
<211> 15
<212> PRT
<213> Homo sapiens
<400> 1981
Ile Pro Asp Trp Ile Val Asp Leu Arg His Glu Leu Thr His Lys
1 5 10 15
<210> 1982
<211> 16
<212> PRT
<213> Thermus thermophilus
<400> 1982
Ile Gln Arg Phe Glu Tyr Thr Phe Glu Ala Phe Trp Lys Ala Leu Gln
1 5 10 15
<210> 1983
<211> 15
<212> PRT
<213> Thermus thermophilus
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<400> 1983
Leu Ala Leu Gly Xaa Val Asp Asp Arg Ser Leu Thr Val His Thr
1 5 10 15
<210> 1984
<211> 16
<212> PRT
<213> Thermus thermophilus
<400> 1984
Ile Gln Arg Phe Glu Tyr Thr Phe Glu Ala Phe Trp Lys Ala Leu Gln
1 5 10 15
<210> 1985
<211> 15
<212> PRT
<213> Thermus thermophilus
<220>
<221> MOD_RES
<222> (5)..(5)
<223> Any amino acid
<400> 1985
Leu Ala Leu Gly Xaa Val Asp Asp Arg Ser Leu Thr Val His Thr
1 5 10 15
<210> 1986
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 1986
Asp Tyr Glu Ile Ile Lys Ala Glu Glu Ser Leu Gln Arg Asp Ile Ser
1 5 10 15
<210> 1987
<211> 15
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(15)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 1987
Leu Lys Ser Met Leu Tyr Ser Met Arg Asn Ser Ser Phe His Phe
1 5 10 15
<210> 1988
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 1988
Leu Ile Lys Trp Cys Tyr Leu Arg Glu Arg Asp Leu Met Tyr Phe Gln
1 5 10 15
<210> 1989
<211> 14
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 1989
Phe Arg Asn Glu Ile Asp His Phe His Tyr Phe Tyr Asp Arg
1 5 10
<210> 1990
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 1990
Asp Tyr Glu Leu Ile Lys Ala Glu Glu Met Leu Gln Arg Glu Thr Ala
1 5 10 15
<210> 1991
<211> 15
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(15)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 1991
Leu Lys Asp Val Ile Tyr Ser Met Arg Asn Asp Ser Phe His Tyr
1 5 10 15
<210> 1992
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(6)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 1992
Leu Ile Asn Trp Ile Tyr Leu Arg Glu Arg Asp Leu Met Asn Phe Gln
1 5 10 15
<210> 1993
<211> 14
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 1993
Leu Arg Asn Tyr Ile Glu His Phe Arg Tyr Tyr Ser Ser Phe
1 5 10
<210> 1994
<211> 16
<212> PRT
<213> Clostridium aminophilum
<400> 1994
Asp Tyr Glu Gln Ile Arg Ala Asp Glu Thr Leu Gln Arg Gln Leu Ser
1 5 10 15
<210> 1995
<211> 15
<212> PRT
<213> Clostridium aminophilum
<400> 1995
Leu Arg Lys Ala Ile Tyr Ser Leu Arg Asn Glu Thr Phe His Phe
1 5 10 15
<210> 1996
<211> 16
<212> PRT
<213> Clostridium aminophilum
<400> 1996
Leu Ile Ser Trp Ser Phe Leu Arg Glu Arg Asp Leu Leu Tyr Phe Gln
1 5 10 15
<210> 1997
<211> 14
<212> PRT
<213> Clostridium aminophilum
<400> 1997
Val Arg Lys Tyr Val Asp His Phe Lys Tyr Tyr Ala Thr Ser
1 5 10
<210> 1998
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 1998
Asp Tyr Glu Gln Ile Lys Ala Asp Glu Met Leu Gln Arg Glu Val Ala
1 5 10 15
<210> 1999
<211> 15
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(15)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 1999
Ile Ile Gln Ile Ile Tyr Ser Leu Arg Asn Lys Ser Phe His Phe
1 5 10 15
<210> 2000
<211> 16
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(16)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 2000
Leu Ile Ser Trp Ser Tyr Leu Arg Glu Arg Asp Leu Leu Tyr Phe Gln
1 5 10 15
<210> 2001
<211> 14
<212> PRT
<213> Unknown
<220>
<223> Synthetic
<220>
<221> source
<222> (1)..(14)
<223> /note="Description of Unknown: Lachnospiraceae bacterium"
<400> 2001
Leu Arg Lys Tyr Val Asp His Phe Lys Tyr Tyr Ala Tyr Gly
1 5 10
<210> 2002
<211> 16
<212> PRT
<213> Carnobacterium gallinarum
<400> 2002
Lys Leu Gln Glu Ile Gly Ile Tyr Glu Gly Phe Gln Thr Lys Phe Met
1 5 10 15
<210> 2003
<211> 15
<212> PRT
<213> Carnobacterium gallinarum
<400> 2003
Leu Arg Gly Ser Val Gln Gln Ile Arg Asn Glu Ile Phe His Ser
1 5 10 15
<210> 2004
<211> 16
<212> PRT
<213> Carnobacterium gallinarum
<400> 2004
Met Val Gly Phe Ser Ala Leu Phe Glu Arg Asp Leu Val Tyr Phe Ser
1 5 10 15
<210> 2005
<211> 14
<212> PRT
<213> Carnobacterium gallinarum
<400> 2005
Ile Arg Asn Gln Thr Ala His Leu Ser Val Leu Gln Leu Glu
1 5 10
<210> 2006
<211> 16
<212> PRT
<213> Carnobacterium gallinarum
<400> 2006
Thr Leu Gln Glu Ile Gly Met Tyr Glu Gly Phe Gln Thr Lys Phe Met
1 5 10 15
<210> 2007
<211> 15
<212> PRT
<213> Carnobacterium gallinarum
<400> 2007
Ile Arg Gly Ala Val Gln Arg Val Arg Asn Gln Ile Phe His Gln
1 5 10 15
<210> 2008
<211> 16
<212> PRT
<213> Carnobacterium gallinarum
<400> 2008
Met Val Gly Phe Ser Val Leu Phe Glu Arg Asp Phe Val Tyr Met Gly
1 5 10 15
<210> 2009
<211> 14
<212> PRT
<213> Carnobacterium gallinarum
<400> 2009
Ile Arg Asn Asn Ile Ala His Leu His Val Leu Arg Asn Asp
1 5 10
<210> 2010
<211> 16
<212> PRT
<213> Paludibacter propionicigenes
<400> 2010
Asp Leu Ile Arg Ile Lys Thr Asn Glu Ala Phe Val Leu Asn Leu Thr
1 5 10 15
<210> 2011
<211> 15
<212> PRT
<213> Paludibacter propionicigenes
<400> 2011
Ile Arg Gly Ala Val Gln Gln Ile Arg Asn Asn Val Asn His Tyr
1 5 10 15
<210> 2012
<211> 16
<212> PRT
<213> Paludibacter propionicigenes
<400> 2012
Met Ala Gly Phe Val Ala Leu Phe Asp Arg Asp Phe Gln Phe Phe Asp
1 5 10 15
<210> 2013
<211> 14
<212> PRT
<213> Paludibacter propionicigenes
<400> 2013
Ile Arg Asn His Ile Ala His Phe Asn Tyr Leu Thr Lys Asp
1 5 10
<210> 2014
<211> 16
<212> PRT
<213> Listeria seeligeri
<400> 2014
Ser Leu Gln Lys Ile Lys Ile Glu Glu Ala Phe Ala Leu Lys Phe Ile
1 5 10 15
<210> 2015
<211> 15
<212> PRT
<213> Listeria seeligeri
<400> 2015
Leu Arg Gly Ala Ile Ala Pro Ile Arg Asn Glu Ile Ile His Leu
1 5 10 15
<210> 2016
<211> 16
<212> PRT
<213> Listeria seeligeri
<400> 2016
Leu Ala Gly Tyr Met Ser Ile Ala Asp Arg Asp Phe Gln Phe Ser Ser
1 5 10 15
<210> 2017
<211> 14
<212> PRT
<213> Listeria seeligeri
<400> 2017
Lys Arg Asn Asn Ile Ser His Phe Asn Tyr Leu Asn Gly Gln
1 5 10
<210> 2018
<211> 16
<212> PRT
<213> Listeria weihenstephanensis
<400> 2018
Asp Leu Gln Asp Ile Arg Ser Gly Glu Ala Phe Ser Phe Lys Phe Ile
1 5 10 15
<210> 2019
<211> 15
<212> PRT
<213> Listeria weihenstephanensis
<400> 2019
Ile Arg Gly Ser Ile Gln Gln Ile Arg Asn Glu Val Tyr His Cys
1 5 10 15
<210> 2020
<211> 16
<212> PRT
<213> Listeria weihenstephanensis
<400> 2020
Tyr Val Gly Tyr Val Ala Ile Ala Asp Arg Asp Phe Gln Cys Met Ala
1 5 10 15
<210> 2021
<211> 14
<212> PRT
<213> Listeria weihenstephanensis
<400> 2021
Ala Arg Asn His Ile Ala His Leu Asn Tyr Leu Ser Leu Lys
1 5 10
<210> 2022
<211> 16
<212> PRT
<213> Listeria newyorkensis
<400> 2022
Asp Leu Gln Asp Ile Arg Ser Gly Glu Ala Phe Ser Phe Lys Phe Ile
1 5 10 15
<210> 2023
<211> 15
<212> PRT
<213> Listeria newyorkensis
<400> 2023
Ile Arg Gly Ser Ile Gln Gln Ile Arg Asn Glu Val Tyr His Cys
1 5 10 15
<210> 2024
<211> 16
<212> PRT
<213> Listeria newyorkensis
<400> 2024
Tyr Val Gly Tyr Val Ala Ile Ala Asp Arg Asp Phe Gln Cys Met Ala
1 5 10 15
<210> 2025
<211> 14
<212> PRT
<213> Listeria newyorkensis
<400> 2025
Ala Arg Asn His Ile Ala His Leu Asn Tyr Leu Ser Leu Lys
1 5 10
<210> 2026
<211> 16
<212> PRT
<213> Leptotrichia wadei
<400> 2026
Asp Leu Glu Tyr Ile Lys Thr Lys Glu Thr Leu Ile Arg Lys Met Ala
1 5 10 15
<210> 2027
<211> 15
<212> PRT
<213> Leptotrichia wadei
<400> 2027
Ile Ser Tyr Ser Ile Tyr Asn Val Arg Asn Gly Val Gly His Phe
1 5 10 15
<210> 2028
<211> 16
<212> PRT
<213> Leptotrichia wadei
<400> 2028
Asn Val Ala Phe Phe Asn Lys Trp Glu Arg Asp Phe Lys Phe Ile Val
1 5 10 15
<210> 2029
<211> 14
<212> PRT
<213> Leptotrichia wadei
<400> 2029
Phe Arg Asn Tyr Ile Ala His Phe Leu His Leu His Thr Lys
1 5 10
<210> 2030
<211> 16
<212> PRT
<213> Leptotrichia wadei
<400> 2030
Ile Asn Met Arg Asn Arg Gln Thr Glu Ala Phe Leu Arg Ser Ile Ile
1 5 10 15
<210> 2031
<211> 15
<212> PRT
<213> Leptotrichia wadei
<400> 2031
Met Leu Asn Ala Ile Thr Ser Ile Arg His Arg Val Val His Tyr
1 5 10 15
<210> 2032
<211> 16
<212> PRT
<213> Leptotrichia wadei
<400> 2032
Leu Ala Gly Tyr Thr Ser Leu Trp Glu Arg Asp Leu Gln Phe Lys Leu
1 5 10 15
<210> 2033
<211> 14
<212> PRT
<213> Leptotrichia wadei
<400> 2033
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro Asp Ala
1 5 10
<210> 2034
<211> 16
<212> PRT
<213> Leptotrichia wadei
<400> 2034
Phe Ile Ala Arg Asn Arg Gln Asn Glu Ala Phe Leu Arg Asn Ile Ile
1 5 10 15
<210> 2035
<211> 15
<212> PRT
<213> Leptotrichia wadei
<400> 2035
Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe
1 5 10 15
<210> 2036
<211> 16
<212> PRT
<213> Leptotrichia wadei
<400> 2036
Leu Val Gly Tyr Thr Ser Ile Trp Glu Arg Asp Leu Arg Phe Arg Leu
1 5 10 15
<210> 2037
<211> 14
<212> PRT
<213> Leptotrichia wadei
<400> 2037
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro His Ala
1 5 10
<210> 2038
<211> 16
<212> PRT
<213> Rhodobacter capsulatus
<400> 2038
Gly Gln Thr Glu Ile Lys Glu Ser Glu Ile Phe Val Arg Leu Trp Val
1 5 10 15
<210> 2039
<211> 15
<212> PRT
<213> Rhodobacter capsulatus
<400> 2039
Leu Leu Arg Tyr Leu Arg Gly Cys Arg Asn Gln Thr Phe His Leu
1 5 10 15
<210> 2040
<211> 16
<212> PRT
<213> Rhodobacter capsulatus
<400> 2040
Leu Ile Asp Tyr Ala Gly Ala Tyr Glu Arg Asp Thr Gly Thr Phe Leu
1 5 10 15
<210> 2041
<211> 14
<212> PRT
<213> Rhodobacter capsulatus
<400> 2041
Thr Arg Lys Asp Leu Ala His Phe Asn Val Leu Asp Arg Ala
1 5 10
<210> 2042
<211> 16
<212> PRT
<213> Leptotrichia buccalis
<400> 2042
Phe Ile Ala Arg Asn Arg Gln Asn Glu Ala Phe Leu Arg Asn Ile Ile
1 5 10 15
<210> 2043
<211> 15
<212> PRT
<213> Leptotrichia buccalis
<400> 2043
Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe
1 5 10 15
<210> 2044
<211> 16
<212> PRT
<213> Leptotrichia buccalis
<400> 2044
Leu Val Gly Tyr Thr Ser Ile Trp Glu Arg Asp Leu Arg Phe Arg Leu
1 5 10 15
<210> 2045
<211> 14
<212> PRT
<213> Leptotrichia buccalis
<400> 2045
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro His Ala
1 5 10
<210> 2046
<211> 16
<212> PRT
<213> Leptotrichia sp.
<400> 2046
Phe Ile Val Gly Asn Arg Gln Asn Glu Ala Phe Leu Arg Asn Ile Ile
1 5 10 15
<210> 2047
<211> 15
<212> PRT
<213> Leptotrichia sp.
<400> 2047
Ile Asp Glu Ala Ile Ser Ser Ile Arg His Gly Ile Val His Phe
1 5 10 15
<210> 2048
<211> 16
<212> PRT
<213> Leptotrichia sp.
<400> 2048
Leu Val Gly Tyr Thr Ser Ile Trp Glu Arg Asp Leu Arg Phe Arg Leu
1 5 10 15
<210> 2049
<211> 14
<212> PRT
<213> Leptotrichia sp.
<400> 2049
Ile Arg Asn Tyr Ile Ala His Phe Asn Tyr Ile Pro Asn Ala
1 5 10
<210> 2050
<211> 16
<212> PRT
<213> Leptotrichia sp.
<400> 2050
Asp Phe Ser Arg Leu His Ala Lys Glu Glu Leu Asp Leu Glu Leu Ile
1 5 10 15
<210> 2051
<211> 15
<212> PRT
<213> Leptotrichia sp.
<400> 2051
Phe Gln Lys Glu Gly Tyr Leu Leu Arg Asn Lys Ile Leu His Asn
1 5 10 15
<210> 2052
<211> 16
<212> PRT
<213> Leptotrichia sp.
<400> 2052
Leu Ala Ile Gln Met Ala Arg Phe Glu Arg Asp Met His Tyr Ile Val
1 5 10 15
<210> 2053
<211> 14
<212> PRT
<213> Leptotrichia sp.
<400> 2053
Ile Arg Asn Tyr Ile Ser His Phe Tyr Ile Val Arg Asn Pro
1 5 10
<210> 2054
<211> 16
<212> PRT
<213> Leptotrichia shahii
<400> 2054
Asp Phe Ser Arg Leu His Ala Lys Glu Glu Leu Asp Leu Glu Leu Ile
1 5 10 15
<210> 2055
<211> 15
<212> PRT
<213> Leptotrichia shahii
<400> 2055
Phe Thr Lys Ile Gly Thr Asn Glu Arg Asn Arg Ile Leu His Ala
1 5 10 15
<210> 2056
<211> 16
<212> PRT
<213> Leptotrichia shahii
<400> 2056
Leu Ala Ile Gln Met Ala Arg Phe Glu Arg Asp Met His Tyr Ile Val
1 5 10 15
<210> 2057
<211> 14
<212> PRT
<213> Leptotrichia shahii
<400> 2057
Ile Arg Asn Tyr Ile Ser His Phe Tyr Ile Val Arg Asn Pro
1 5 10
<210> 2058
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2058
ccaccccaau aucgaagggg acuaaaacaa guuugcagcu ggauacgaca gacggc 56
<210> 2059
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2059
ccaccccaau aucgaagggg acuaaaacaa guuugcagcu ggauacgaca gacggc 56
<210> 2060
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2060
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 2061
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2061
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 2062
<211> 173
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2062
ggccagugaa uucgagcucg guacccgggg auccucuaga aauauggauu acuugguaga 60
acagcaaucu acucgaccug caggcaugca agcuuggcgu aaucaugguc auagcuguuu 120
ccuguguuua uccgcucaca auuccacaca acauacgagc cggaagcaua aag 173
<210> 2063
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2063
uagauugcug uucuaccaag uaauccau 28
<210> 2064
<211> 176
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2064
ggguaggugu uccacagggu agccagcagc auccugcgau gcaaauaugg auuacuuggu 60
agaacagcaa ucuaauccgg aacauaaugg ugcagggcgc ugacuuccgc guuuccagac 120
uuuacgaaac acggaaaccg aagaccauuc auguuguugc ugccggaagc auaaag 176
<210> 2065
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2065
uagauugcug uucuaccaag uaauccau 28
<210> 2066
<211> 176
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2066
gggccccucc guucgcguuu acgcggacgg ugagacugaa gauaauaugg auuacuuggu 60
agaacagcaa ucuaaacuca uucucuuuaa aauaucguuc gaacuggacu cccggucguu 120
uuaacucgac uggggccaaa acgaaacagu ggcacuaccc cgccggaagc auaaag 176
<210> 2067
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2067
uagauugcug uucuaccaag uaauccau 28
<210> 2068
<211> 173
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2068
ggccagugaa uucgagcucg guacccgggg auccucuaga aauauggauu acuugguaga 60
acagcaaucu acucgaccug caggcaugca agcuuggcgu aaucaugguc auagcuguuu 120
ccuguguuua uccgcucaca auuccacaca acauacgagc cggaagcaua aag 173
<210> 2069
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2069
uagauugcug uucuaccaag uaauccau 28
<210> 2070
<211> 176
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2070
gggccccucc guucgcguuu acgcggacgg ugagacugaa gauaauaugg auuacuuggu 60
agaacagcaa ucuaaacuca uucucuuuaa aauaucguuc gaacuggacu cccggucguu 120
uuaacucgac uggggccaaa acgaaacagu ggcacuaccc cgccggaagc auaaag 176
<210> 2071
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2071
uagauugcug uucuaccaag uaauccau 28
<210> 2072
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2072
gtcaggtcgg tactaacatc aagttagatg gccgtctgtc 40
<210> 2073
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2073
gacagacggc catctaactt gatgttag 28
<210> 2074
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2074
gacggccatc taacttgatg ttagtacc 28
<210> 2075
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2075
gccatctaac ttgatgttag taccgacc 28
<210> 2076
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2076
tctaacttga tgttagtacc gacctgac 28
<210> 2077
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2077
ccgucugucg uauccagcug caaacuucc 29
<210> 2078
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2078
gucguaucca gcugcaaacu uccagacaa 29
<210> 2079
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2079
guuagauggc cgucugucgu auccagcug 29
<210> 2080
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2080
cagacaacgu gcaacauauc gcgacguau 29
<210> 2081
<211> 70
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2081
uacccgggga uccucuagaa auauggauua cuugguagaa cagcaaucua cucgaccugc 60
aggcaugcaa 70
<210> 2082
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2082
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 2083
<211> 125
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2083
gagcucggua cccggggauc cucuagaaau auggauuacu ugguagaaca gcaaucuacu 60
cgaccugcag gcaugcaagc uuggcguaau cauggucaua gcuguuuccu guguuuaucc 120
gcuca 125
<210> 2084
<211> 173
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2084
ggccagugaa uucgagcucg guacccgggg auccucuaga aauauggauu acuugguaga 60
acagcaaucu acucgaccug caggcaugca agcuuggcgu aaucaugguc auagcuguuu 120
ccuguguuua uccgcucaca auuccacaca acauacgagc cggaagcaua aag 173
<210> 2085
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2085
uagauugcug uucuaccaag uaauccau 28
<210> 2086
<211> 176
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2086
ggguaggugu uccacagggu agccagcagc auccugcgau gcaaauaugg auuacuuggu 60
agaacagcaa ucuaauccgg aacauaaugg ugcagggcgc ugacuuccgc guuuccagac 120
uuuacgaaac acggaaaccg aagaccauuc auguuguugc ugccggaagc auaaag 176
<210> 2087
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2087
uagauugcug uucuaccaag uaauccau 28
<210> 2088
<211> 176
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2088
gggccccucc guucgcguuu acgcggacgg ugagacugaa gauaauaugg auuacuuggu 60
agaacagcaa ucuaaacuca uucucuuuaa aauaucguuc gaacuggacu cccggucguu 120
uuaacucgac uggggccaaa acgaaacagu ggcacuaccc cgccggaagc auaaag 176
<210> 2089
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2089
uagauugcug uucuaccaag uaauccau 28
<210> 2090
<211> 55
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2090
aucaaguuag auggccgucu gucguaucca gcugcaaacu uccagacaac gugca 55
<210> 2091
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2091
ccaccccaau aucgaagggg acuaaaacaa guuugcagcu ggauacgaca gacggc 56
<210> 2092
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2092
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 2093
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2093
uagauugcug uucuaccaag uaau 24
<210> 2094
<211> 23
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2094
uagauugcug uucuaccaag uaa 23
<210> 2095
<211> 12
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2095
uagauugcug uu 12
<210> 2096
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2096
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 2097
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2097
ccacccgaau aucgaacggg acuaaaac 28
<210> 2098
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2098
ccaccgcaau aucgaagcgg acuaaaac 28
<210> 2099
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2099
ccacgccaau aucgaaggcg acuaaaac 28
<210> 2100
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2100
ccagcccaau aucgaagggc acuaaaac 28
<210> 2101
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2101
ccaggggaau aucgaacccc acuaaaac 28
<210> 2102
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2102
ccaccaccaa uaucgaaggg gacuaaaac 29
<210> 2103
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2103
ccaacccaau aucgaagggg acuaaaac 28
<210> 2104
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2104
ccacccaaau aucgaagggg acuaaaac 28
<210> 2105
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2105
ccacccccaa uaucgaaggg ggacuaaaac 30
<210> 2106
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2106
ccaccccaau aucgaagggg acuaaaac 28
<210> 2107
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2107
ccaccccaua ucgaagggga cuaaaac 27
<210> 2108
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2108
ccaccccauc gaaggggacu aaaac 25
<210> 2109
<211> 21
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2109
ccaccccaag gggacuaaaa c 21
<210> 2110
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2110
ccaccccaaa uaucgaaggg gacuaaaac 29
<210> 2111
<211> 31
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2111
ccaccccaaa aauaucgaag gggacuaaaa c 31
<210> 2112
<211> 35
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2112
ccaccccaaa aaaaaauauc gaaggggacu aaaac 35
<210> 2113
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2113
ccaccccgau aucgaagggg acuaaaac 28
<210> 2114
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2114
ccaccccaaa aucgaagggg acuaaaac 28
<210> 2115
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2115
ccaccccaau auccaagggg acuaaaac 28
<210> 2116
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2116
ccaccccaau aucgaagggg acuaaaacaa guuugcagcu ggauacgaca gacggc 56
<210> 2117
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2117
uaguuugcag cuggauacga cagacggc 28
<210> 2118
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2118
aaguaugcag cuggauacga cagacggc 28
<210> 2119
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2119
aaguuugcug cuggauacga cagacggc 28
<210> 2120
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2120
aaguuugcag cucgauacga cagacggc 28
<210> 2121
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2121
aaguuugcag cuggauucga cagacggc 28
<210> 2122
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2122
aaguuugcag cuggauacga gagacggc 28
<210> 2123
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2123
aaguuugcag cuggauacga cagagggc 28
<210> 2124
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2124
ccaccccaau aucgaagggg acuaaaacaa guuugcagcu ggauacgaca gacggc 56
<210> 2125
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2125
uuguuugcag cuggauacga cagacggc 28
<210> 2126
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2126
aaguuaccag cuggauacga cagacggc 28
<210> 2127
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2127
aaguuugcag gaggauacga cagacggc 28
<210> 2128
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2128
aaguuugcag cuggaaucga cagacggc 28
<210> 2129
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2129
aaguuugcag cuggauacga gcgacggc 28
<210> 2130
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2130
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 2131
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2131
aagauugcug uucuaccaag uaauccau 28
<210> 2132
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2132
uagaaugcug uucuaccaag uaauccau 28
<210> 2133
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2133
uagauugcag uucuaccaag uaauccau 28
<210> 2134
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2134
uagauugcug uuguaccaag uaauccau 28
<210> 2135
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2135
uagauugcug uucuacgaag uaauccau 28
<210> 2136
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2136
uagauugcug uucuaccaag aaauccau 28
<210> 2137
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2137
uagauugcug uucuaccaag uaaugcau 28
<210> 2138
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2138
uagauugcug uucuaccaag uaauccaa 28
<210> 2139
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2139
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 2140
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2140
augauugcug uucuaccaag uaauccau 28
<210> 2141
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2141
uagauaccug uucuaccaag uaauccau 28
<210> 2142
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2142
uagauugcug aacuaccaag uaauccau 28
<210> 2143
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2143
uagauugcug uuguaggaag uaauccau 28
<210> 2144
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2144
uagauugcug uucuaccaag auauccau 28
<210> 2145
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2145
uagauugcug uucuaccaag uaaucguu 28
<210> 2146
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2146
uucgaaaucg aaggugaagg ugaaggucgu ccguacgaag guacccagac cgcuaaacug 60
<210> 2147
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2147
aaacuguccc uuccggaagg uuucaaaugg gaacguguua ugaacuucga agacgguggu 60
<210> 2148
<211> 60
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2148
ccaggacucc ucccugcaag acggugaguu caucuacaaa guuaaacugc gugguaccaa 60
<210> 2149
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2149
ccaccccaau aucgaagggg acuaaaac 28
<210> 2150
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<220>
<221> misc_feature
<222> (1)..(56)
<223> /note="Variant nucleotides given in the sequence have no
preference with respect to those in the annotations for variant
positions"
<220>
<221> modified_base
<222> (29)..(56)
<223> a, c, u, g, unknown or other
<220>
<221> variation
<222> (43)..(56)
<223> /replace=" "
<400> 2150
ccaccccaau aucgaagggg acuaaaacnn nnnnnnnnnn nnnnnnnnnn nnnnnn 56
<210> 2151
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2151
cgccuuacaa gaaguucgcu gaacaagca 29
<210> 2152
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2152
uucgcuacga auuccgacug cgagcuuau 29
<210> 2153
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2153
acgggugagu ccaucauaag cguugacgc 29
<210> 2154
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2154
aaccguuuua caucaagaaa ccuguugac 29
<210> 2155
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2155
gccgucuguc guauccagcu gcaaacuuc 29
<210> 2156
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2156
ccgucugucg uauccagcug caaacuucc 29
<210> 2157
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2157
auggccgucu gucguaucca gcugcaaac 29
<210> 2158
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2158
uuagauggcc gucugucgua uccagcugc 29
<210> 2159
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2159
guuagauggc cgucugucgu auccagcug 29
<210> 2160
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2160
cguauccagc ugcaaacuuc cagacaacg 29
<210> 2161
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2161
aacuuccaga caacgugcaa cauaucgcg 29
<210> 2162
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2162
uauccagcug caaacuucca gacaacgug 29
<210> 2163
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2163
gucguaucca gcugcaaacu uccagacaa 29
<210> 2164
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2164
cugcaaacuu ccagacaacg ugcaacaua 29
<210> 2165
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2165
agauggccgu cugucguauc cagcugcaa 29
<210> 2166
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2166
ucugucguau ccagcugcaa acuuccaga 29
<210> 2167
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2167
ggccgucugu cguauccagc ugcaaacuu 29
<210> 2168
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2168
cagacaacgu gcaacauauc gcgacguau 29
<210> 2169
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2169
aguuagaugg ccgucugucg uauccagcu 29
<210> 2170
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2170
uggccgucug ucguauccag cugcaaacu 29
<210> 2171
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2171
ccgucugucg uauccagcug caaacuucc 29
<210> 2172
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2172
gccgucuguc guauccagcu gcaaacuuc 29
<210> 2173
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2173
uaguuugcag cuggauacga cagacggc 28
<210> 2174
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2174
aaguaugcag cuggauacga cagacggc 28
<210> 2175
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2175
aaguuugcug cuggauacga cagacggc 28
<210> 2176
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2176
aaguuugcag cucgauacga cagacggc 28
<210> 2177
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2177
aaguuugcag cuggauucga cagacggc 28
<210> 2178
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2178
aaguuugcag cuggauacga gagacggc 28
<210> 2179
<211> 29
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2179
aaaguuugca gcuggauacg acagagggc 29
<210> 2180
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2180
uuguuugcag cuggauacga cagacggc 28
<210> 2181
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2181
aaguuaccag cuggauacga cagacggc 28
<210> 2182
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2182
aaguuugcag gaggauacga cagacggc 28
<210> 2183
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2183
aaguuugcag cuggaaucga cagacggc 28
<210> 2184
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2184
aaguuugcag cuggauacga gcgacggc 28
<210> 2185
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2185
aaguuugcag cuggauacga cagacggc 28
<210> 2186
<211> 55
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2186
aucaaguuag gguccgcugc cguucgcuug ggacauccug uccagacaac gugca 55
<210> 2187
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2187
ccaccccaau aucgaagggg acuaaaacac aggauguccc aagcgaacgg cagcgg 56
<210> 2188
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2188
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 2189
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2189
aagaaugcug uucuaccaag uaauccau 28
<210> 2190
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2190
aagauugcag uucuaccaag uaauccau 28
<210> 2191
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2191
aagauugcug uuguaccaag uaauccau 28
<210> 2192
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2192
uagaaugcag uucuaccaag uaauccau 28
<210> 2193
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2193
uagaaugcug uuguaccaag uaauccau 28
<210> 2194
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2194
uagaaugcug uucuacgaag uaauccau 28
<210> 2195
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2195
uagauugcag uuguaccaag uaauccau 28
<210> 2196
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2196
uagauugcag uucuacgaag uaauccau 28
<210> 2197
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2197
uagauugcag uuguaccaag aaauccau 28
<210> 2198
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2198
uagauugcug uuguacgaag uaauccau 28
<210> 2199
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2199
uagauugcug uuguaccaag aaauccau 28
<210> 2200
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2200
uagauugcug uuguaccaag uaaugcau 28
<210> 2201
<211> 56
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2201
ccaccccaau aucgaagggg acuaaaacua gauugcuguu cuaccaagua auccau 56
<210> 2202
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2202
aucauugcug uucuaccaag uaauccau 28
<210> 2203
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2203
uagauacgug uucuaccaag uaauccau 28
<210> 2204
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2204
uagauugcug aaguaccaag uaauccau 28
<210> 2205
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2205
uagauugcug uuguagguag uaauccau 28
<210> 2206
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2206
uagauugcug uucuaccaag auuuccau 28
<210> 2207
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2207
uagauugcug uucuaccaag uaaucgua 28
<210> 2208
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2208
ccaccccaau aucgaagggg ucuaaaac 28
<210> 2209
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2209
ccaccccaau aucgaagggg aguaaaac 28
<210> 2210
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2210
ccaccccaau aucgaagggg cuaaaac 27
<210> 2211
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2211
ccaccccaau aucgaagggg auaaaac 27
<210> 2212
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2212
ccaccccaau aucgaagggg acaaaaac 28
<210> 2213
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2213
ccaccccaau aucgaagggg acuuaaac 28
<210> 2214
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2214
ccaccccaau aucgaagggg acaaaac 27
<210> 2215
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2215
ccaccccaau aucgaagggg acuaaac 27
<210> 2216
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2216
ccaccccaau aucgaagggg acuauaac 28
<210> 2217
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2217
ccaccccaau aucgaagggg acuaauac 28
<210> 2218
<211> 26
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2218
ccaccccaau aucgaagggg acuaac 26
<210> 2219
<211> 25
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2219
ccaccccaau aucgaagggg acuac 25
<210> 2220
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2220
ccaccccaau aucgaagggg acuaaauc 28
<210> 2221
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2221
ccaccccaau aucgaagggg acuaaaag 28
<210> 2222
<211> 24
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2222
ccaccccaau aucgaagggg acuc 24
<210> 2223
<211> 27
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2223
ccaccccaau aucgaagggg acuaaaa 27
<210> 2224
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2224
ccaccccaau aucgaagggg acuaaaac 28
<210> 2225
<211> 173
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2225
ggccagugaa uucgagcucg guacccgggg auccucuaga aauauggauu acuugguaga 60
acagcaaucu acucgaccug caggcaugca agcuuggcgu aaucaugguc auagcuguuu 120
ccuguguuua uccgcucaca auuccacaca acauacgagc cggaagcaua aag 173
<210> 2226
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2226
uagauugcug uucuaccaag uaauccau 28
<210> 2227
<211> 176
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2227
gggccccucc guucgcguuu acgcggacgg ugagacugaa gauaauaugg auuacuuggu 60
agaacagcaa ucuaaacuca uucucuuuaa aauaucguuc gaacuggacu cccggucguu 120
uuaacucgac uggggccaaa acgaaacagu ggcacuaccc cgccggaagc auaaag 176
<210> 2228
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2228
uagauugcug uucuaccaag uaauccau 28
<210> 2229
<211> 35
<212> DNA
<213> Alicyclobacillus acidoterrestris
<400> 2229
gtcggatcac tgagagcgga tctgagaagt ggcac 35
<210> 2230
<211> 37
<212> DNA
<213> Desulfonatronum thiodismutans
<400> 2230
gtctcggcaa gcttcgtcag tgttgggtgt atggcac 37
<210> 2231
<211> 30
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2231
ccacccgcaa uaucgaagcg ggacuaaaac 30
<210> 2232
<211> 28
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2232
ccacccaaua aucgaagggg acuaaaac 28
<210> 2233
<211> 78
<212> RNA
<213> Artificial Sequence
<220>
<223> Synthetic
<400> 2233
auuauuacca uuuugguugg aaugcuauua uaaaggauca uucgauuauu accucuaccu 60
cccuucccac gauuucuu 78
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2777988C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2737537C2 |
| ВАКЦИНА И СПОСОБЫ ОБНАРУЖЕНИЯ И ПРОФИЛАКТИКИ ФИЛЯРИОЗА | 2021 |
|
RU2832185C1 |
| T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, РАСПОЗНАЮЩИЕ МУТАЦИЮ R175H ИЛИ Y220C В P53 | 2020 |
|
RU2830061C2 |
| НЕ ВСТРЕЧАЮЩИЙСЯ В ПРИРОДЕ ВИРУС РЕПРОДУКТИВНО-РЕСПИРАТОРНОГО СИНДРОМА СВИНЕЙ (BPPCC) И СПОСОБЫ ПРИМЕНЕНИЯ | 2015 |
|
RU2687150C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2023 |
|
RU2832308C2 |
| ЛЕЧЕНИЕ ЗЛОКАЧЕСТВЕННОГО НОВООБРАЗОВАНИЯ С ИСПОЛЬЗОВАНИЕМ ГУМАНИЗИРОВАННОГО ХИМЕРНОГО АНТИГЕННОГО РЕЦЕПТОРА ПРОТИВ ВСМА | 2015 |
|
RU2751660C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЕГРАДАЦИИ НЕПРАВИЛЬНО УПАКОВАННЫХ БЕЛКОВ | 2016 |
|
RU2761564C2 |
| МАТЕРИАЛЫ И МЕТОДЫ, ИСПОЛЬЗУЕМЫЕ ДЛЯ ЛЕЧЕНИЯ РЕСПИРАТОРНЫХ ЗАБОЛЕВАНИЙ У СОБАК | 2020 |
|
RU2811752C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ ИЗБИРАТЕЛЬНОЙ ЭКСПРЕССИИ БЕЛКА | 2017 |
|
RU2795467C2 |
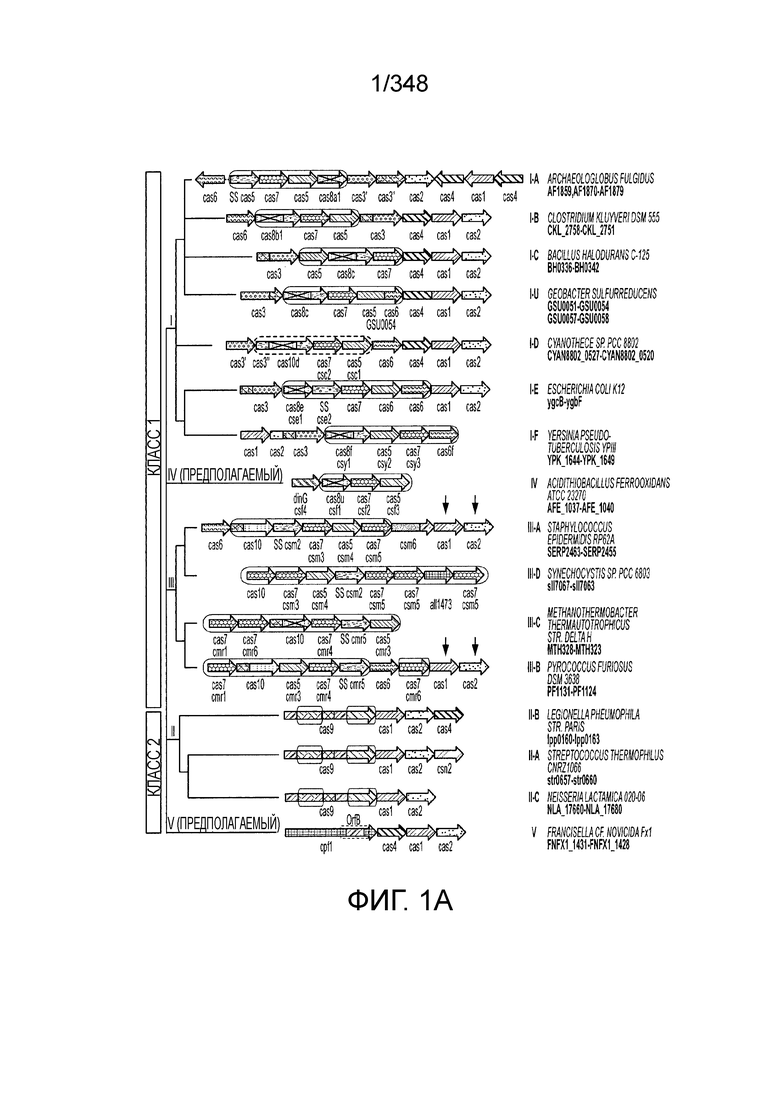
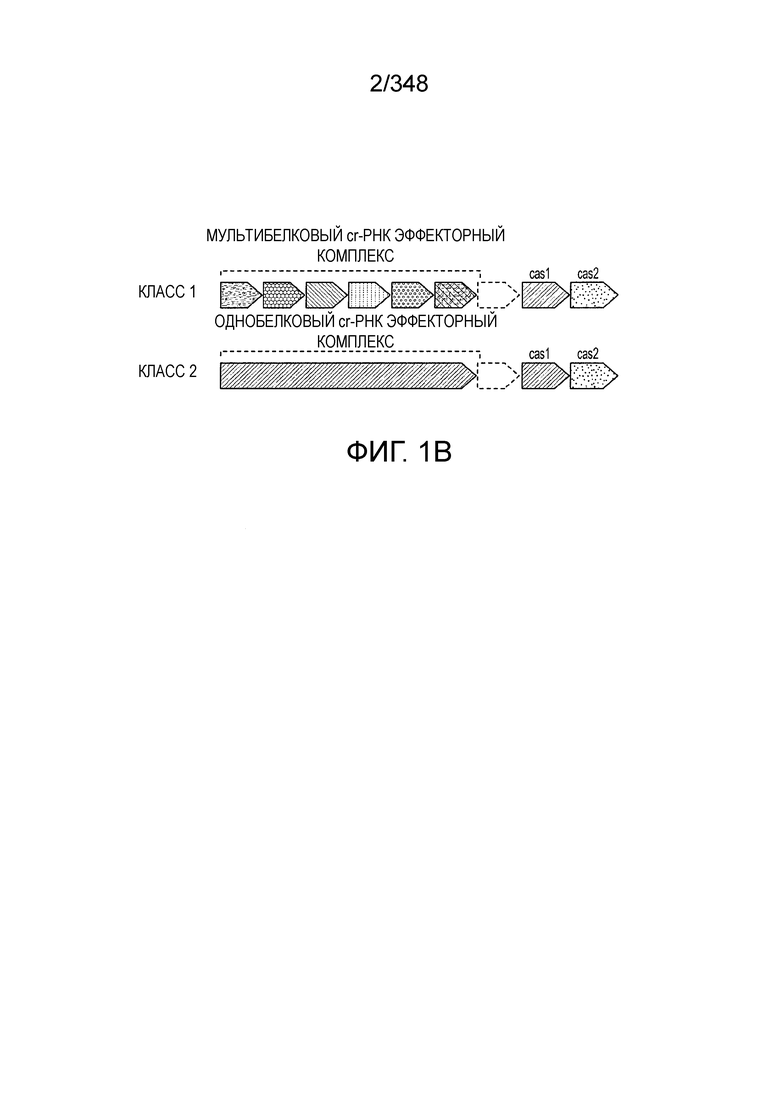
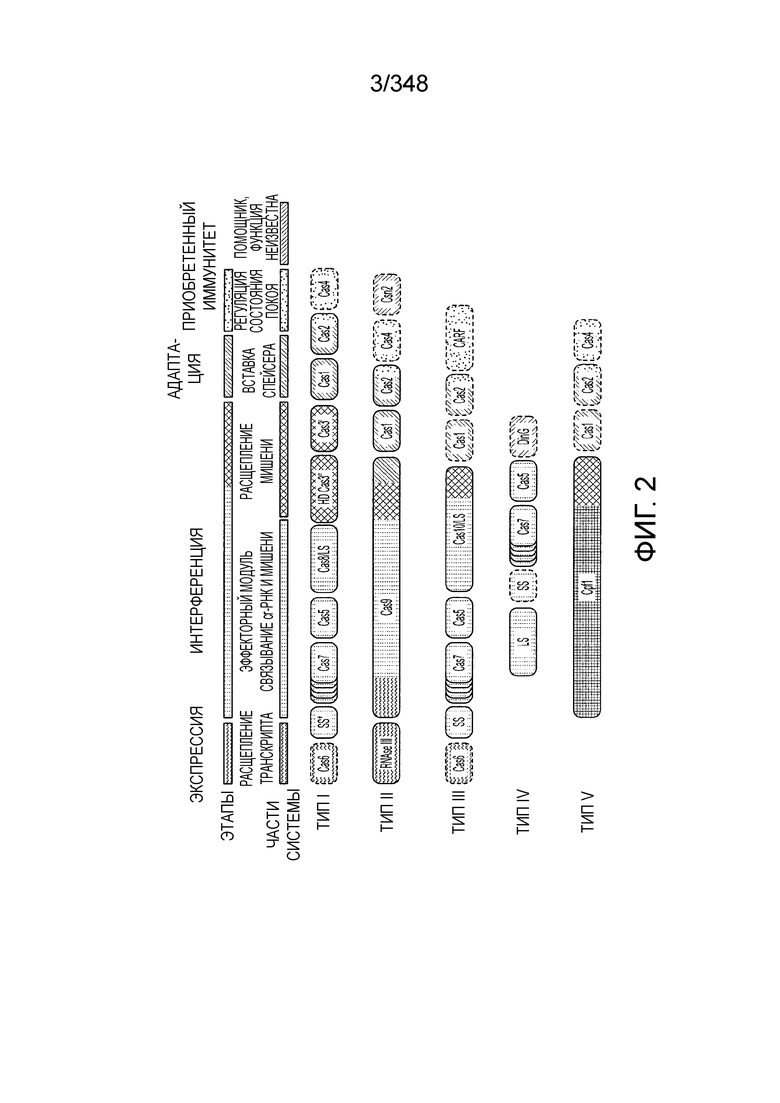
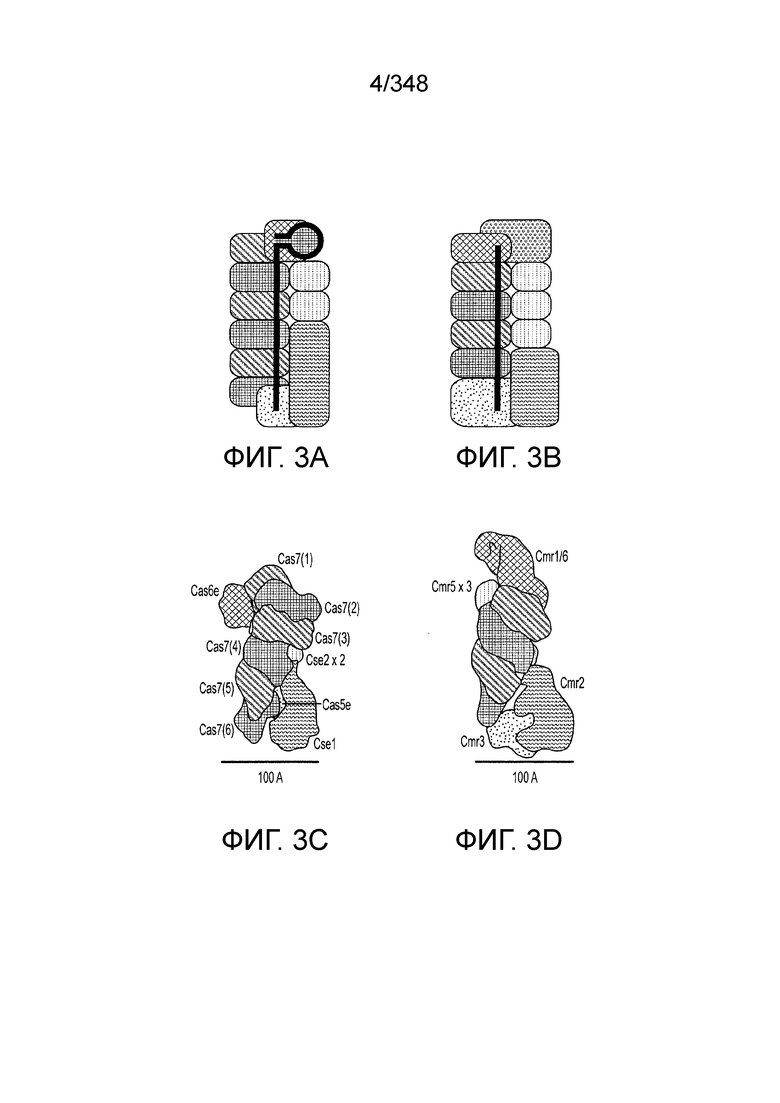
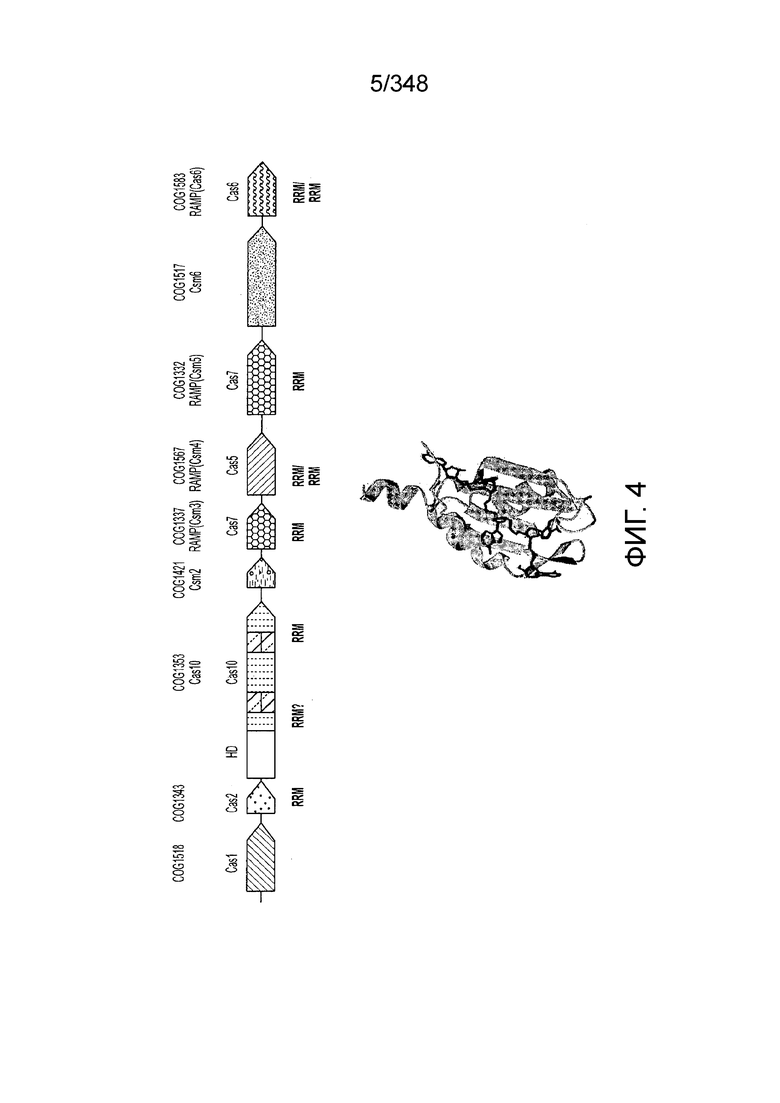
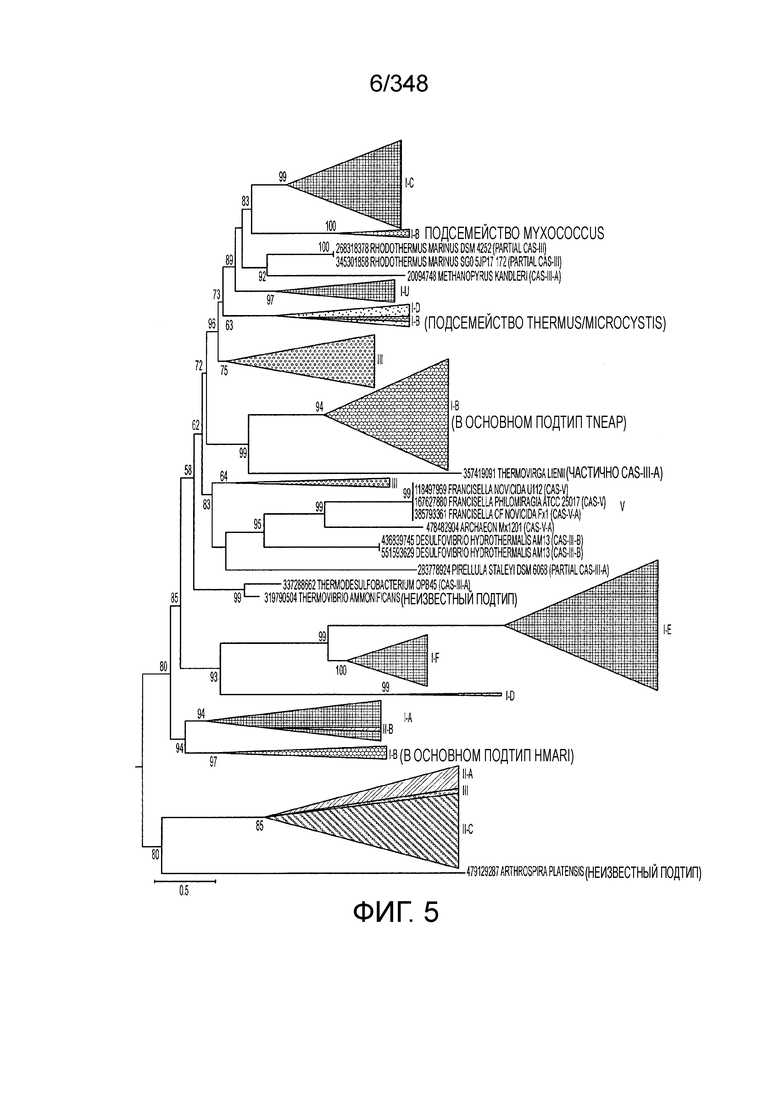

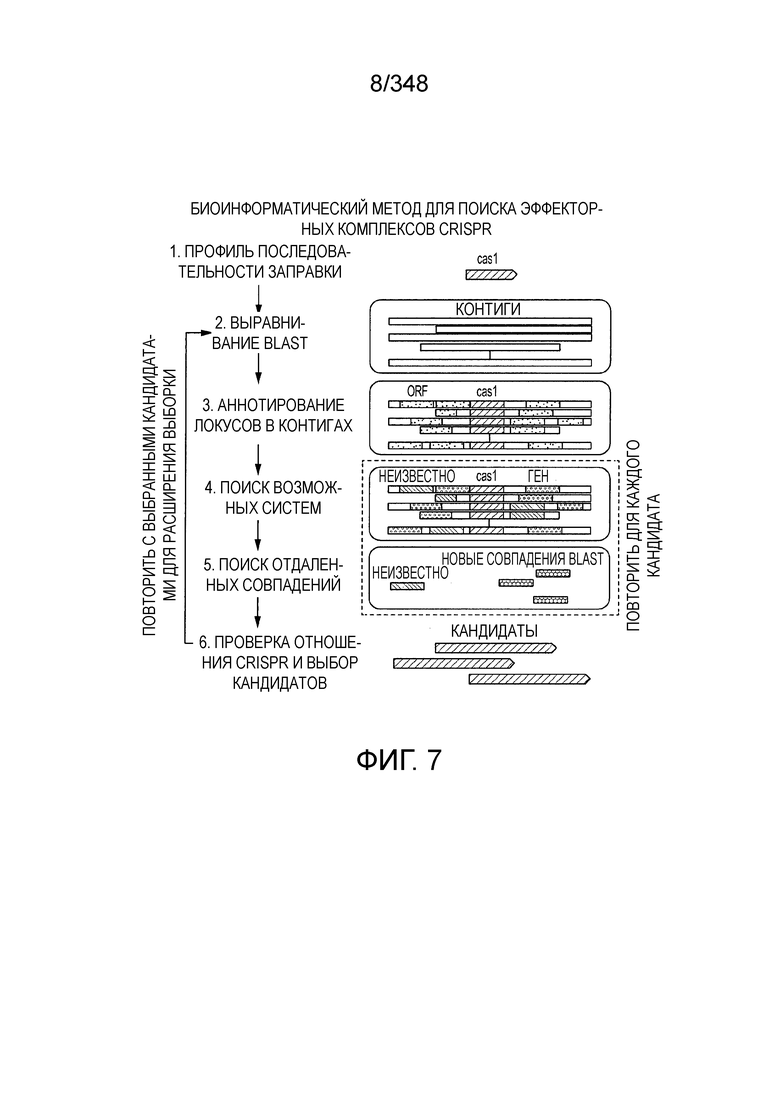
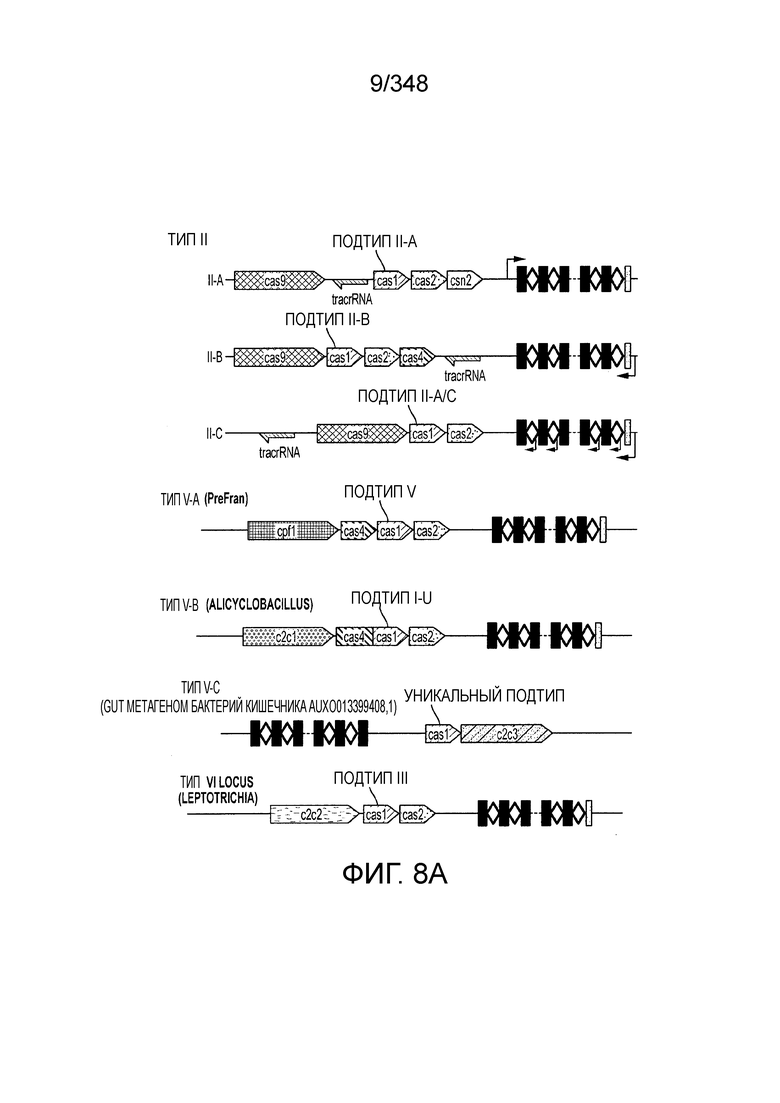
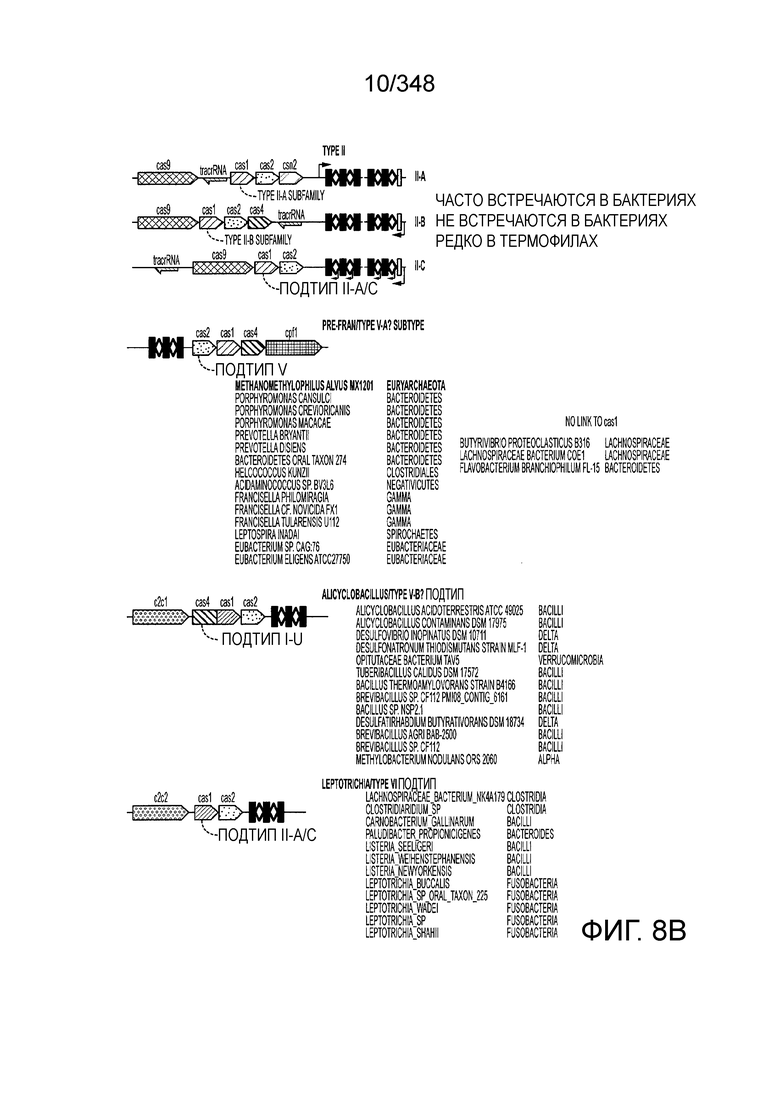
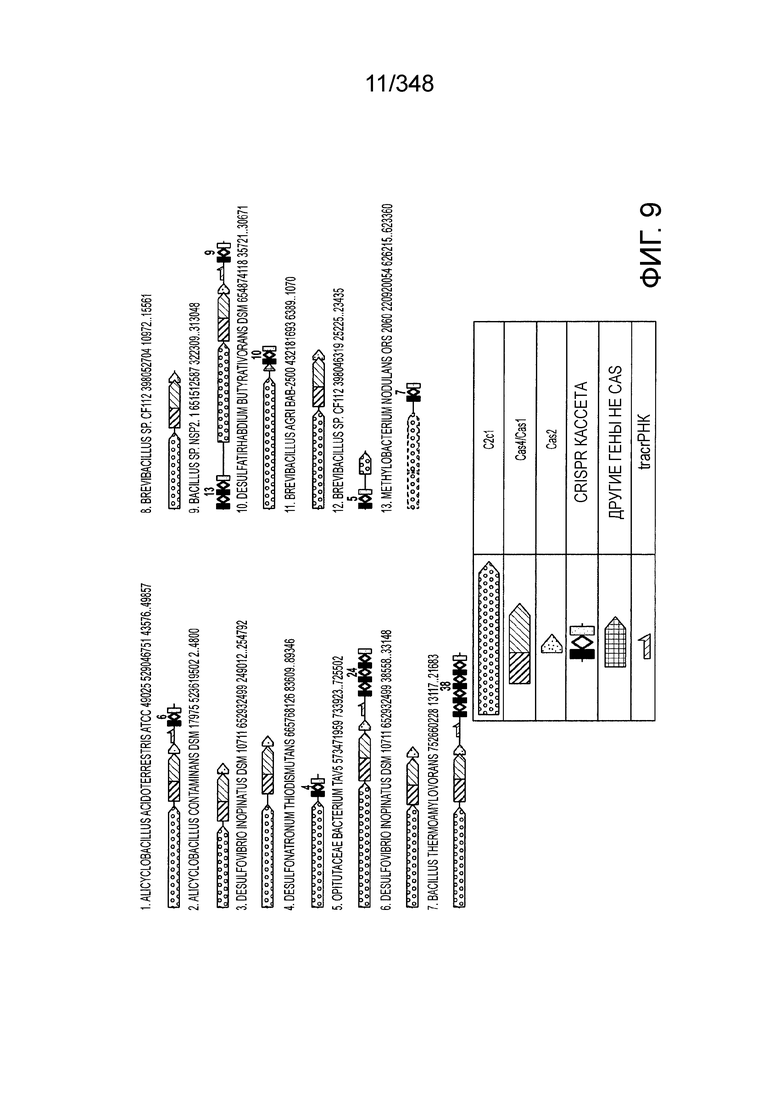

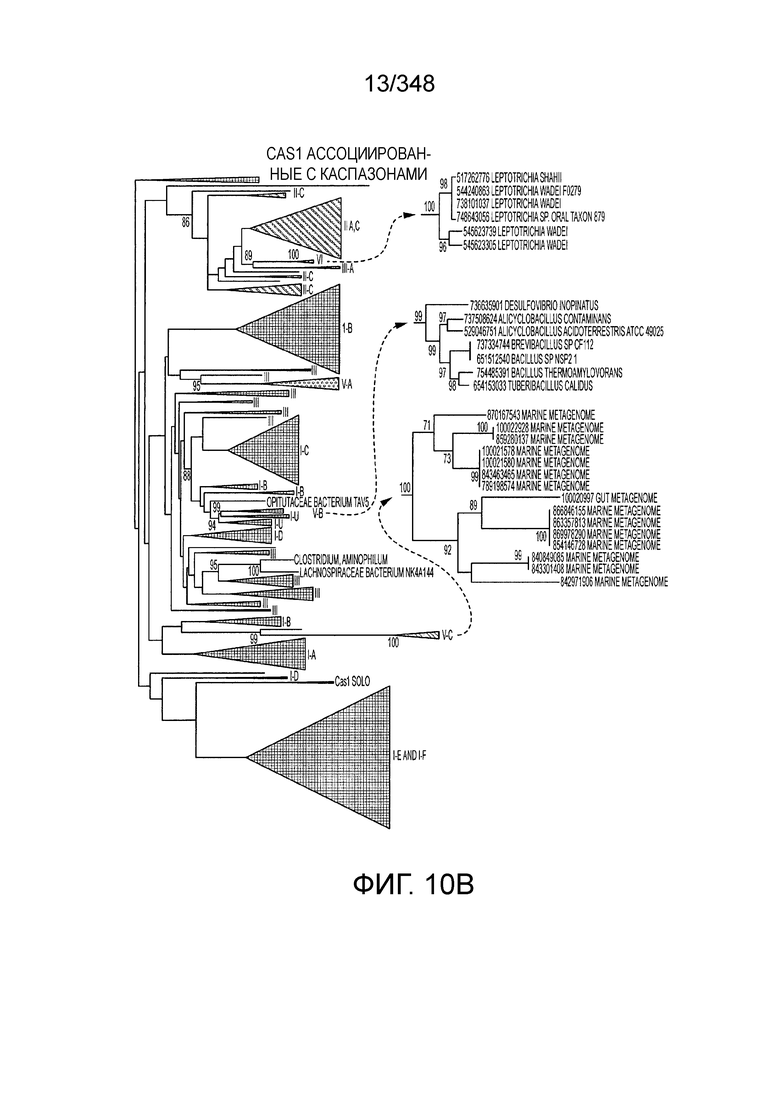









































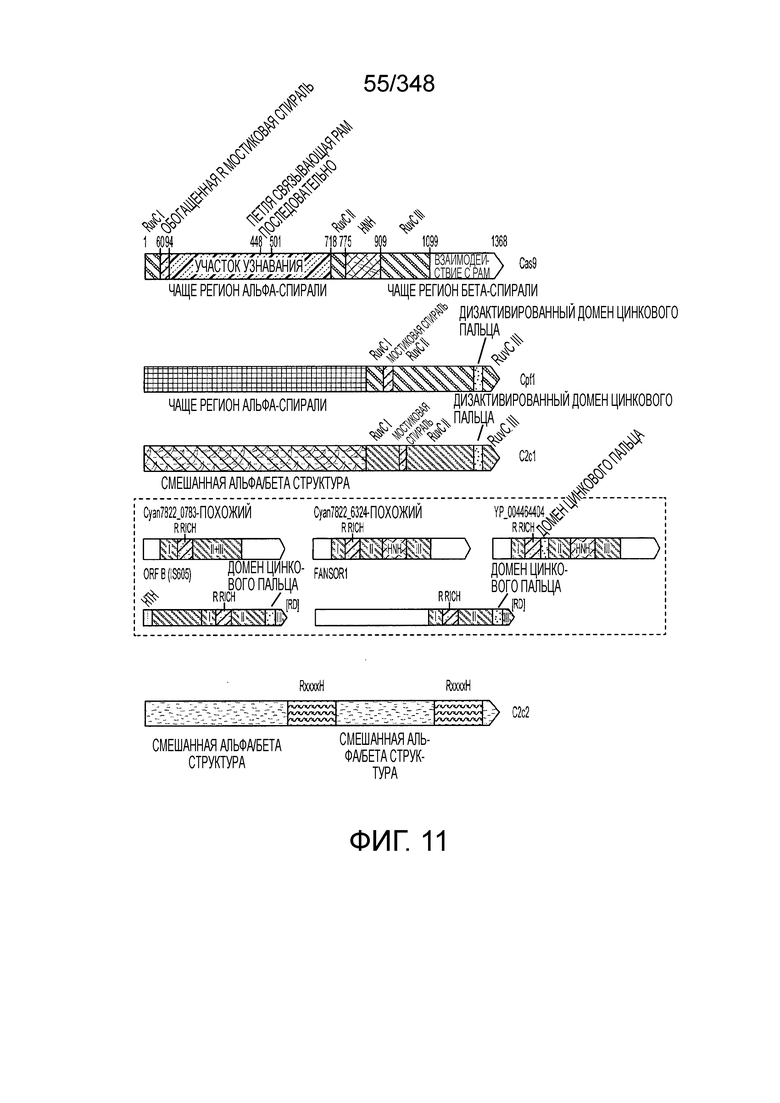

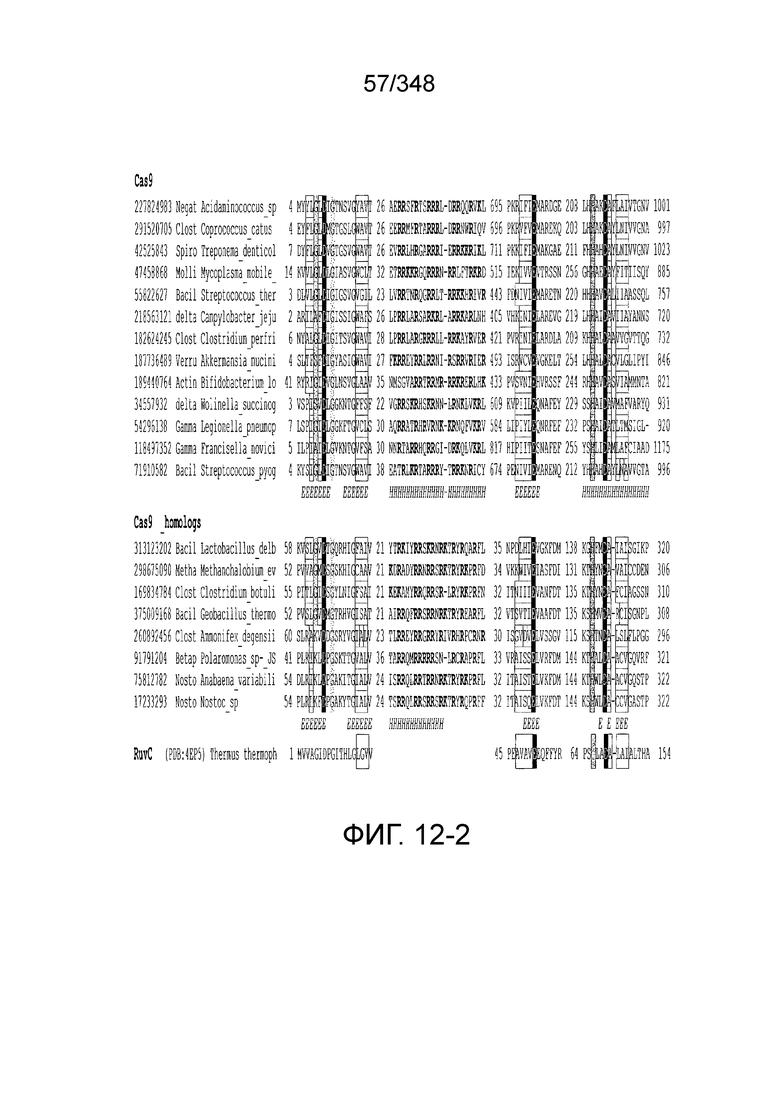

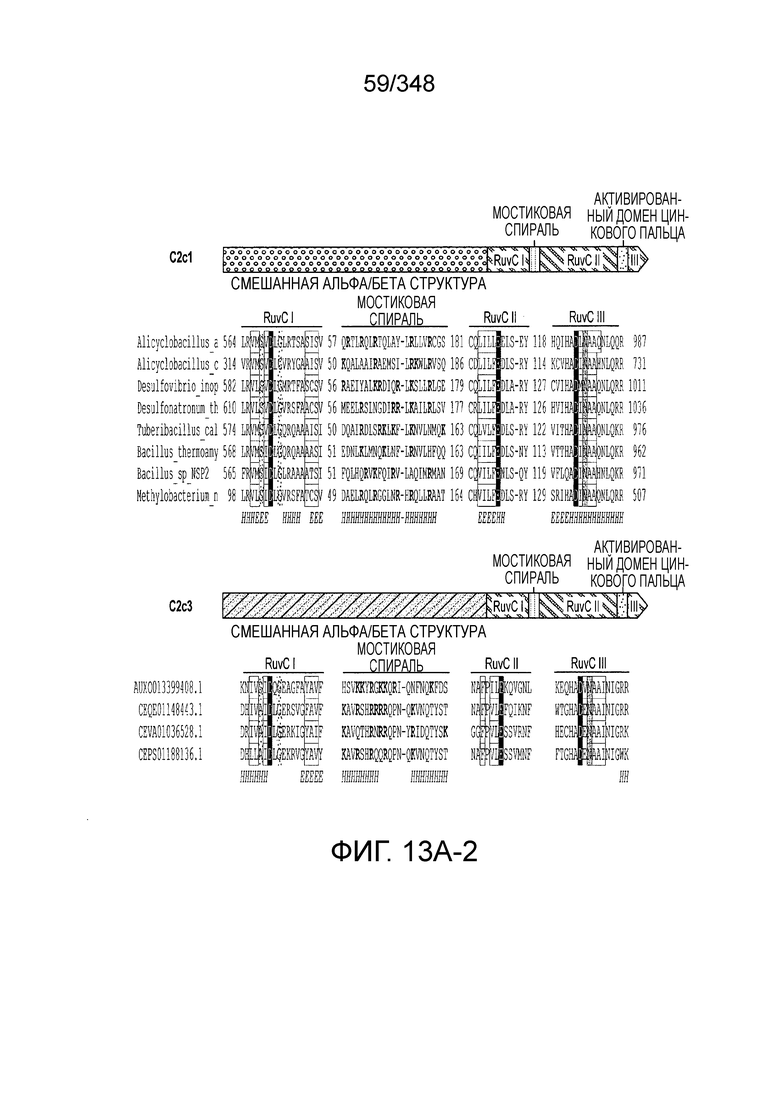
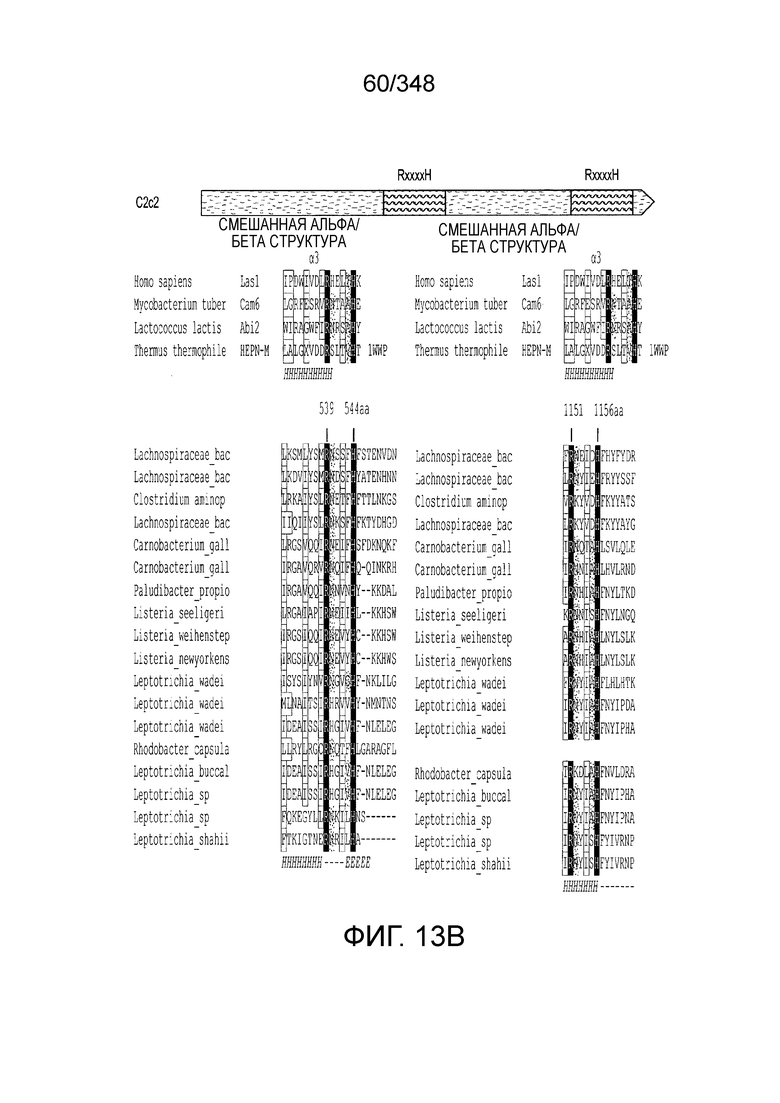
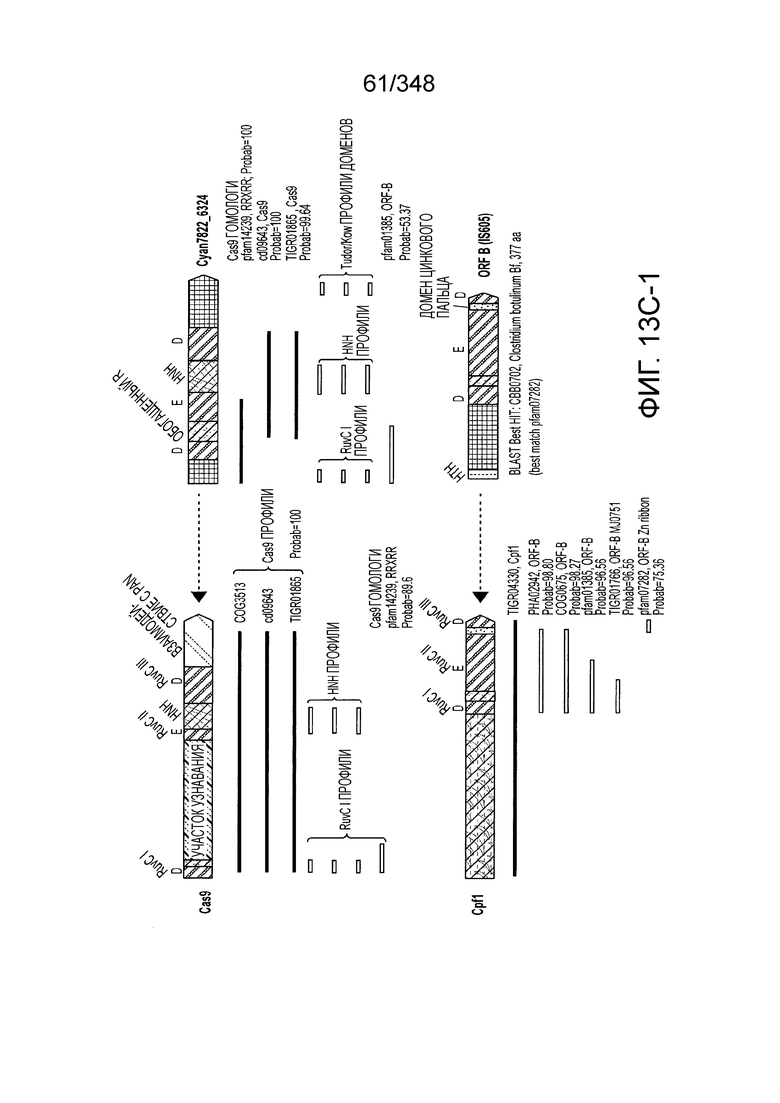
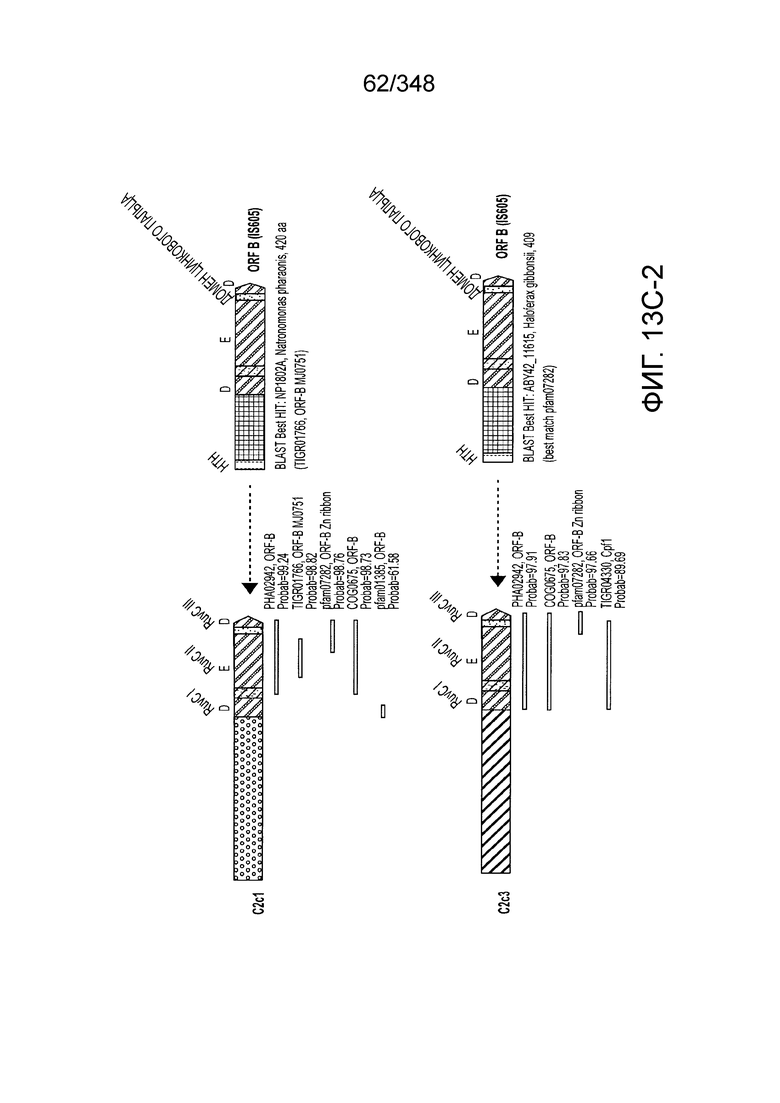








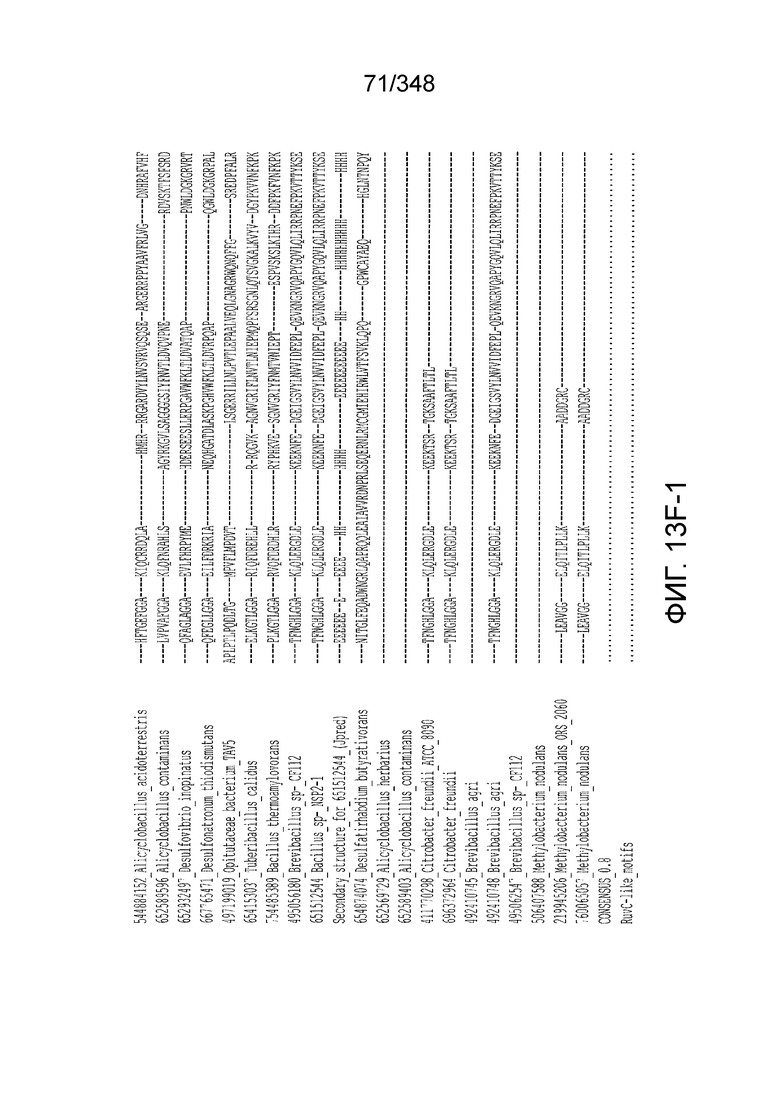



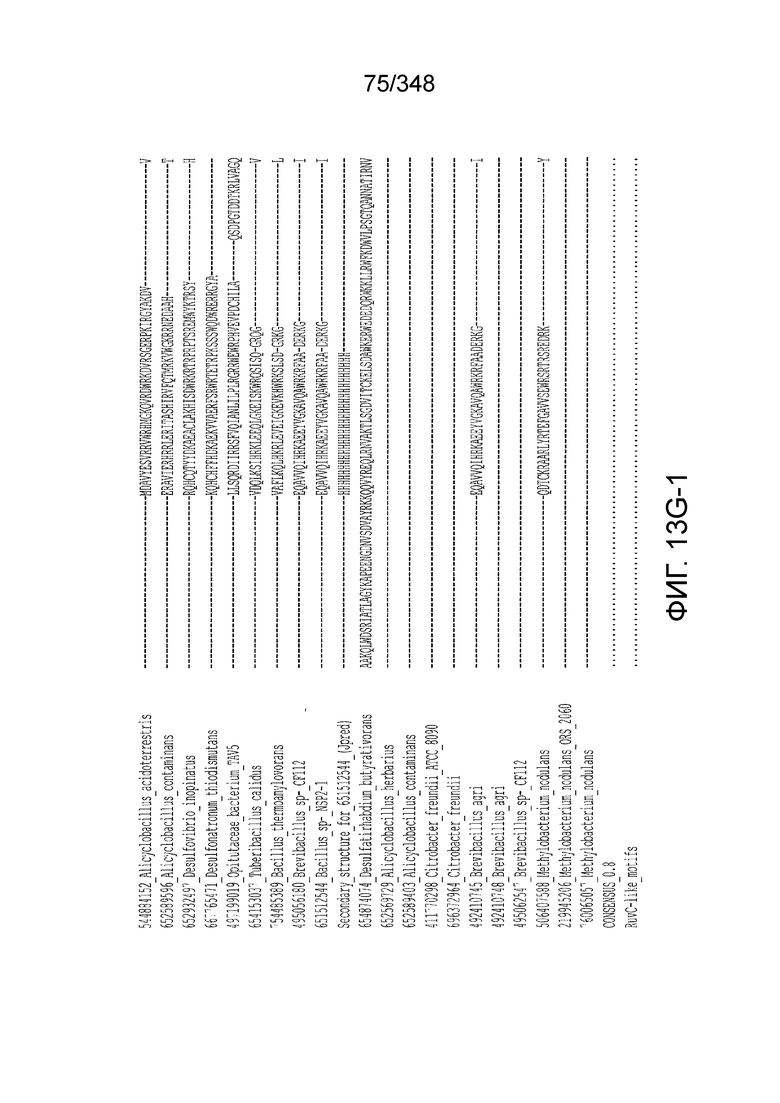


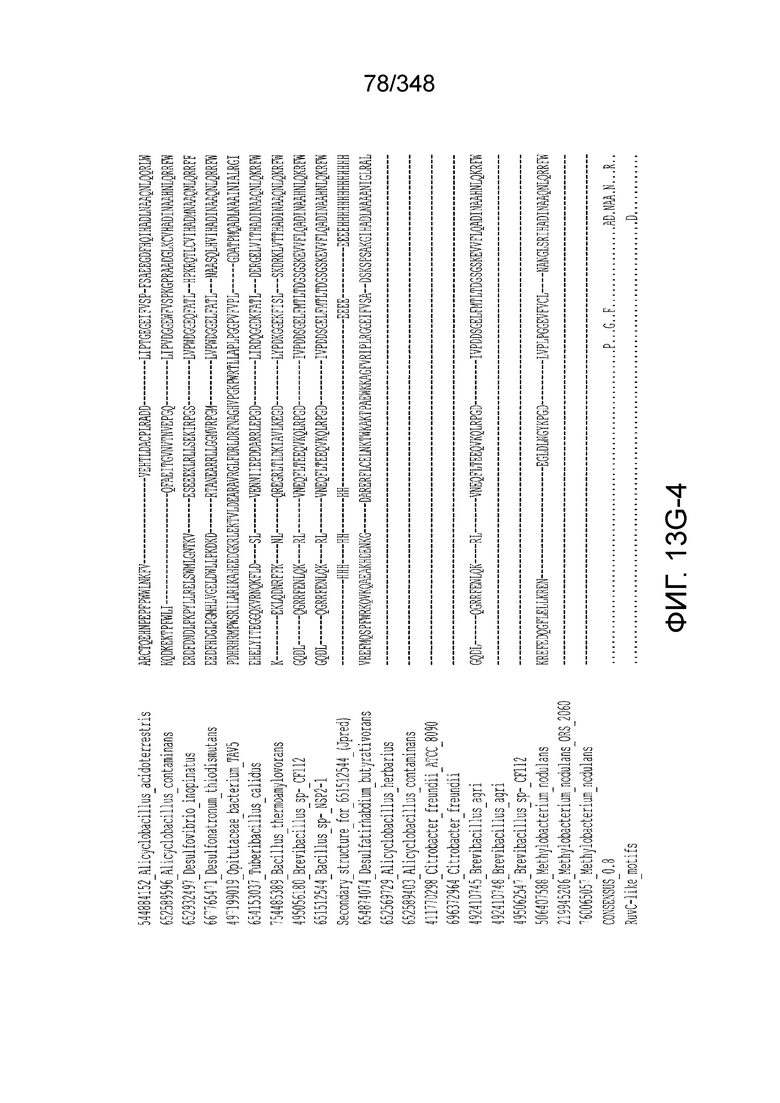


















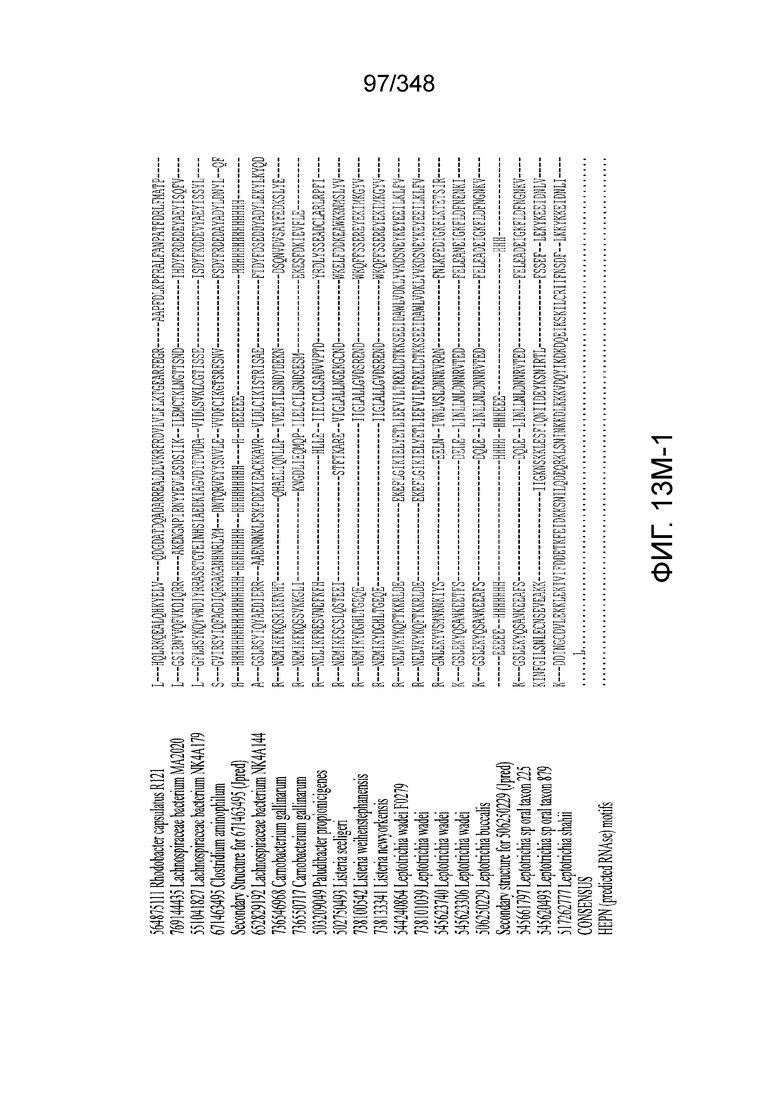
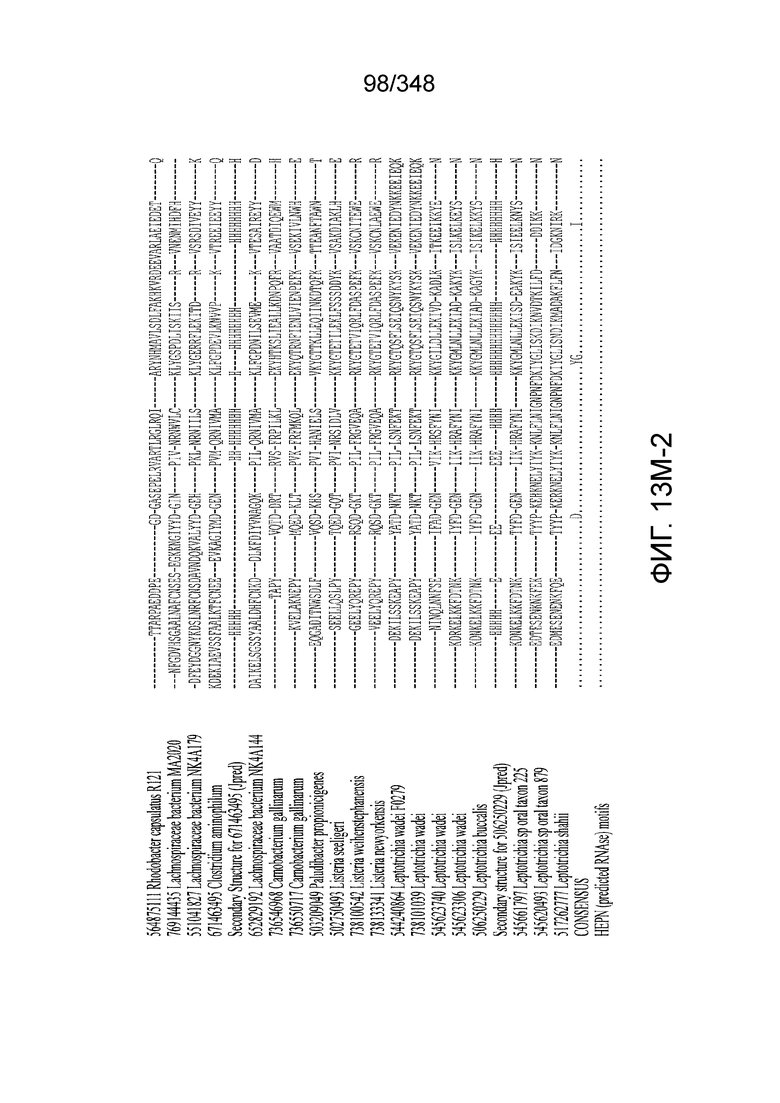



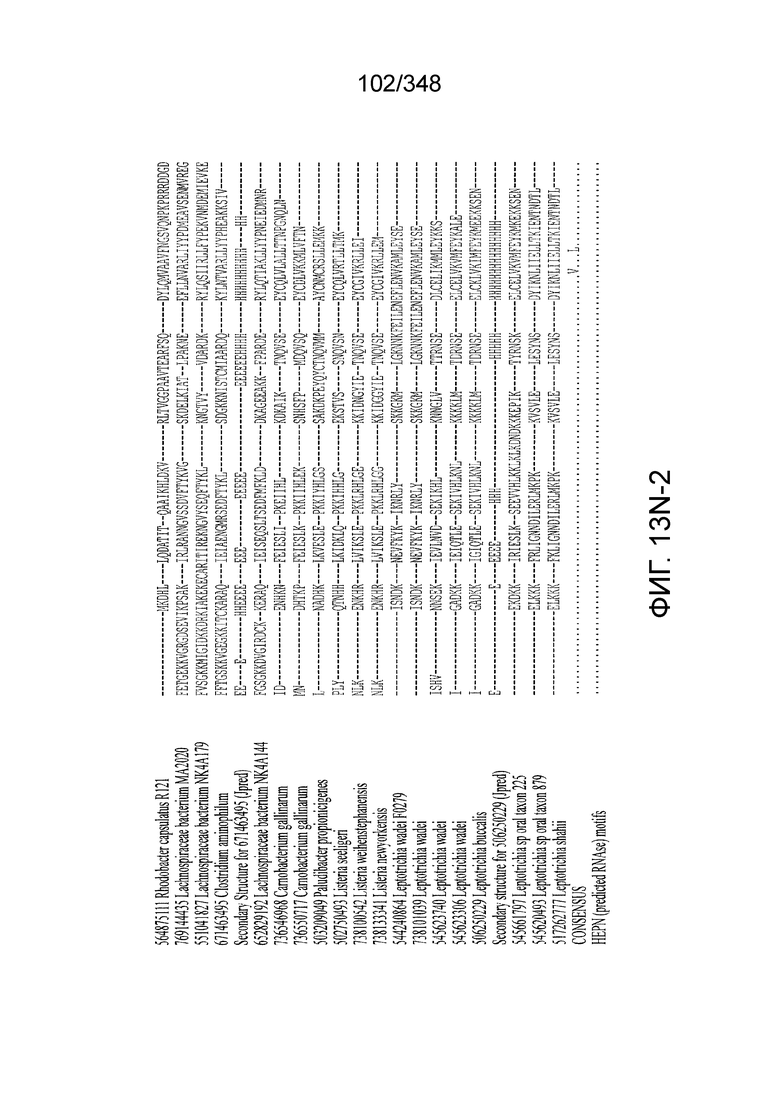



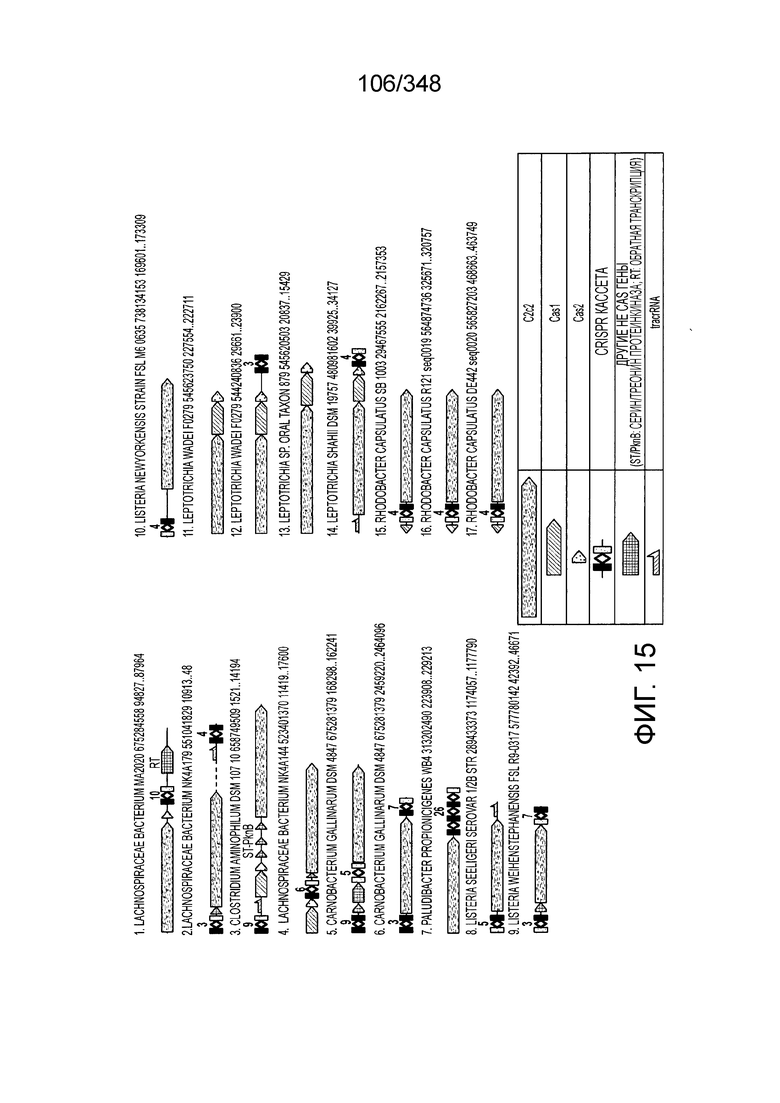





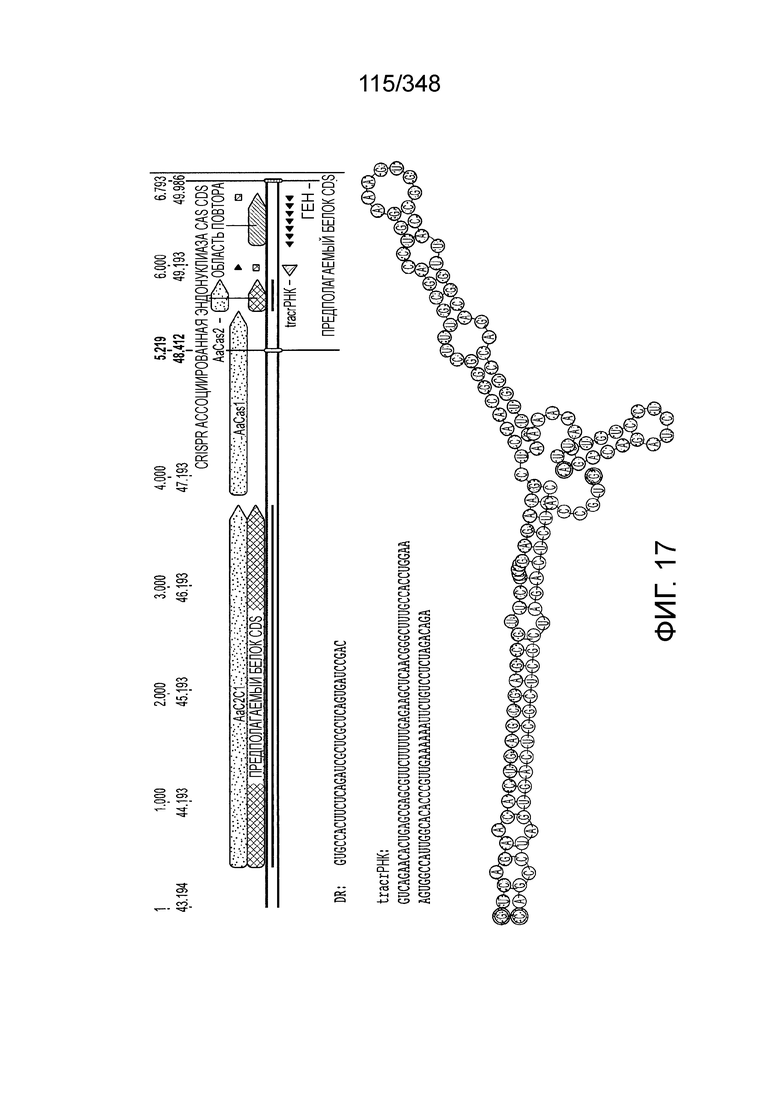
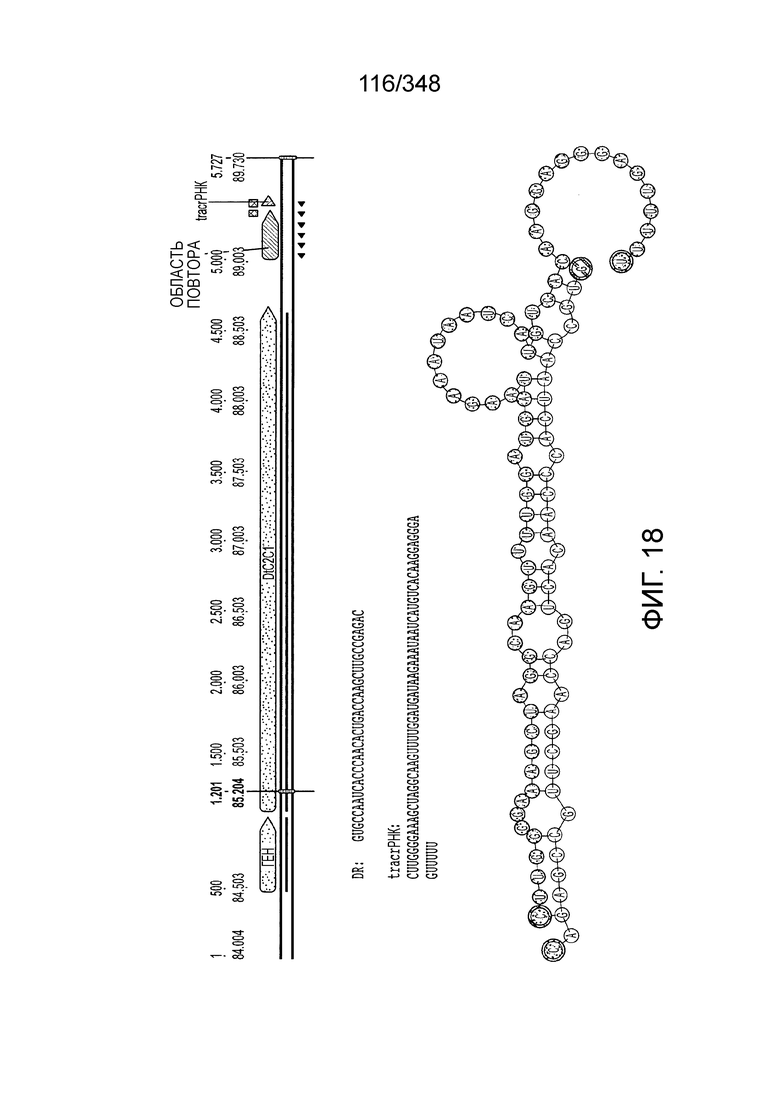
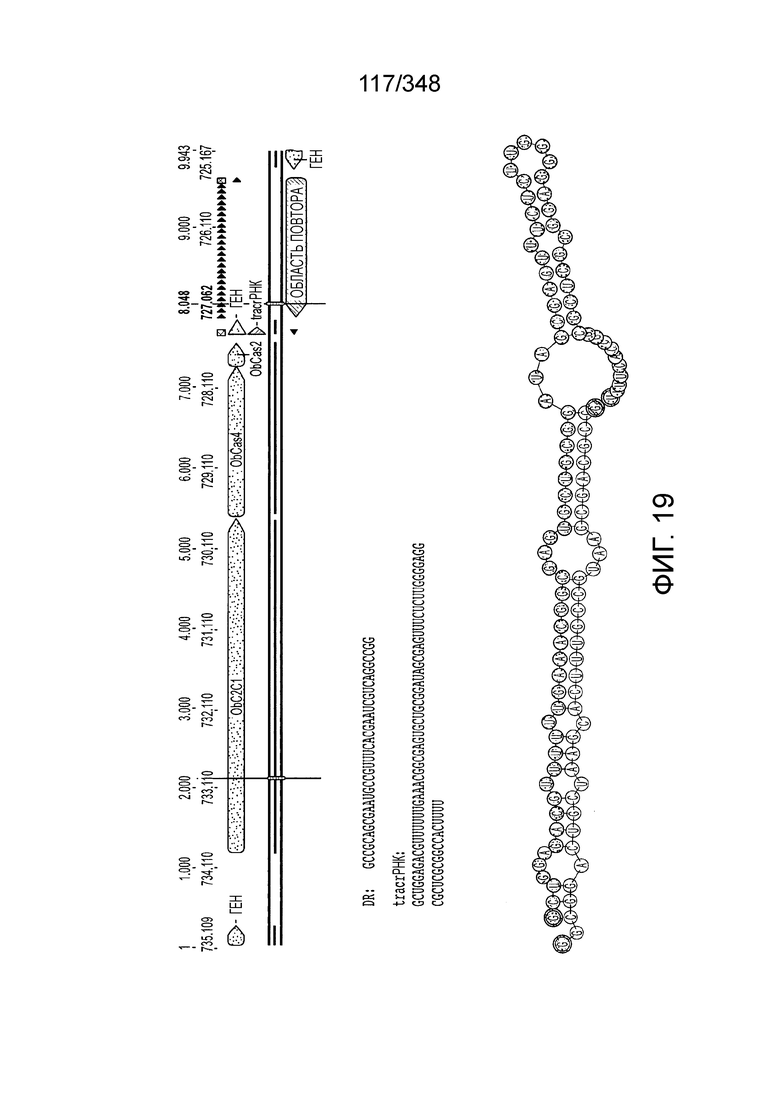
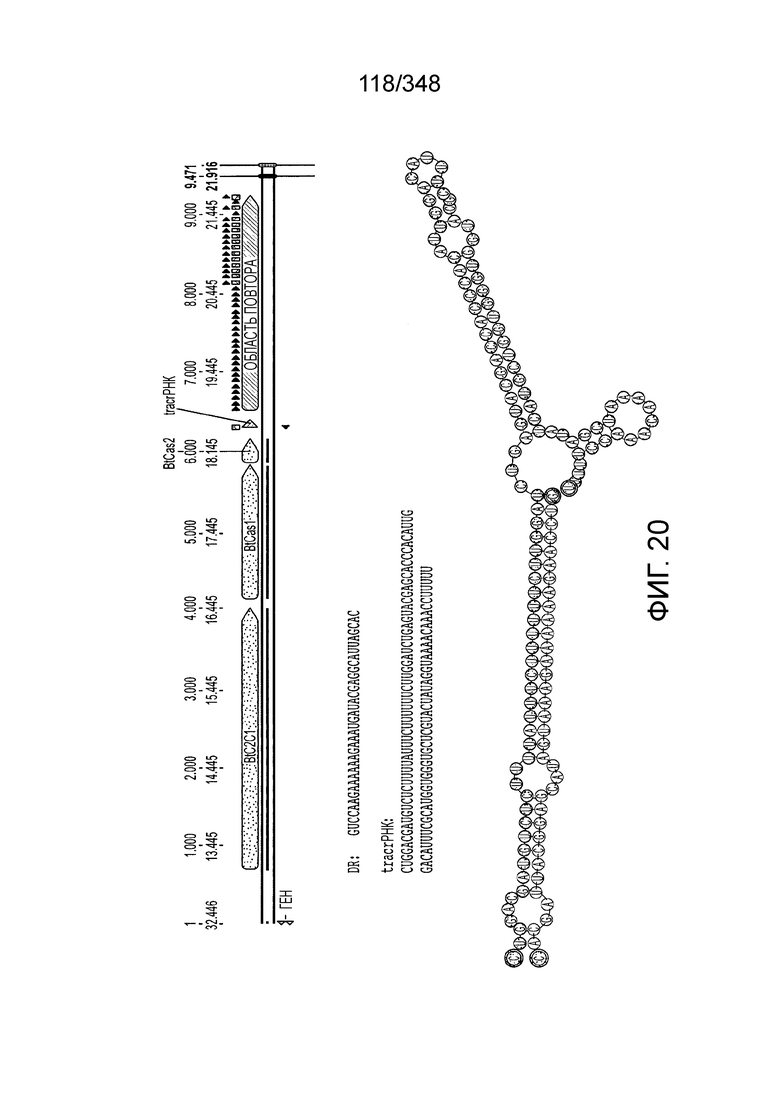
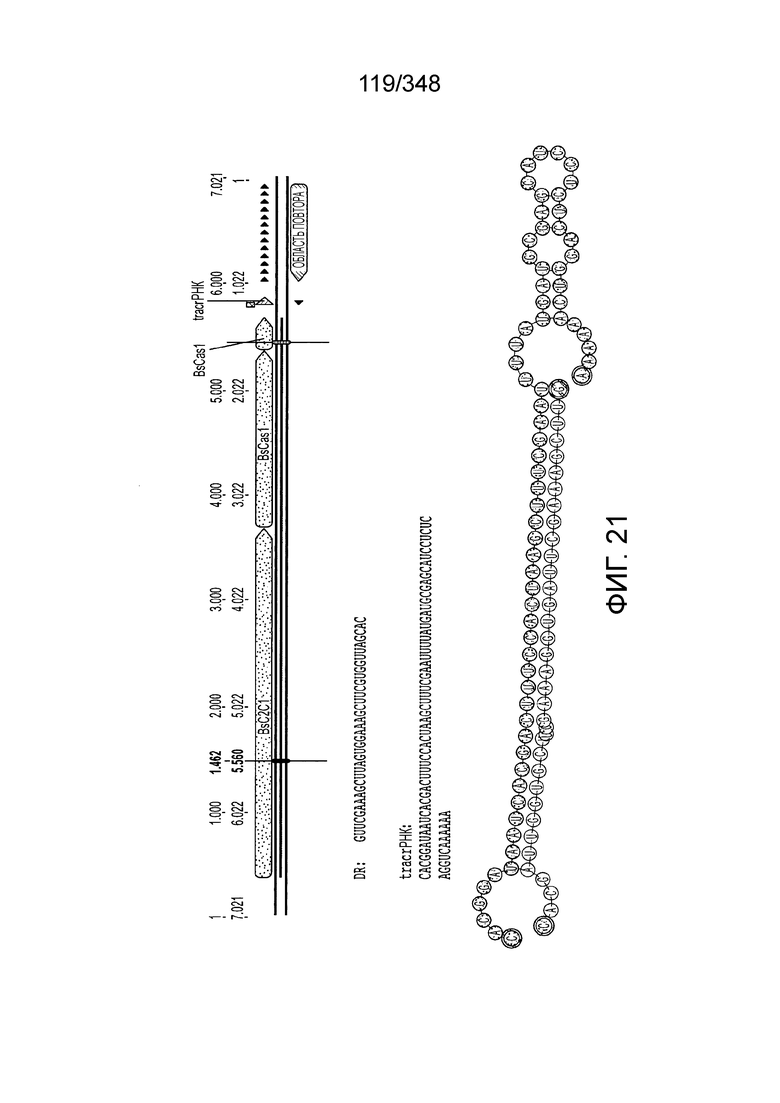
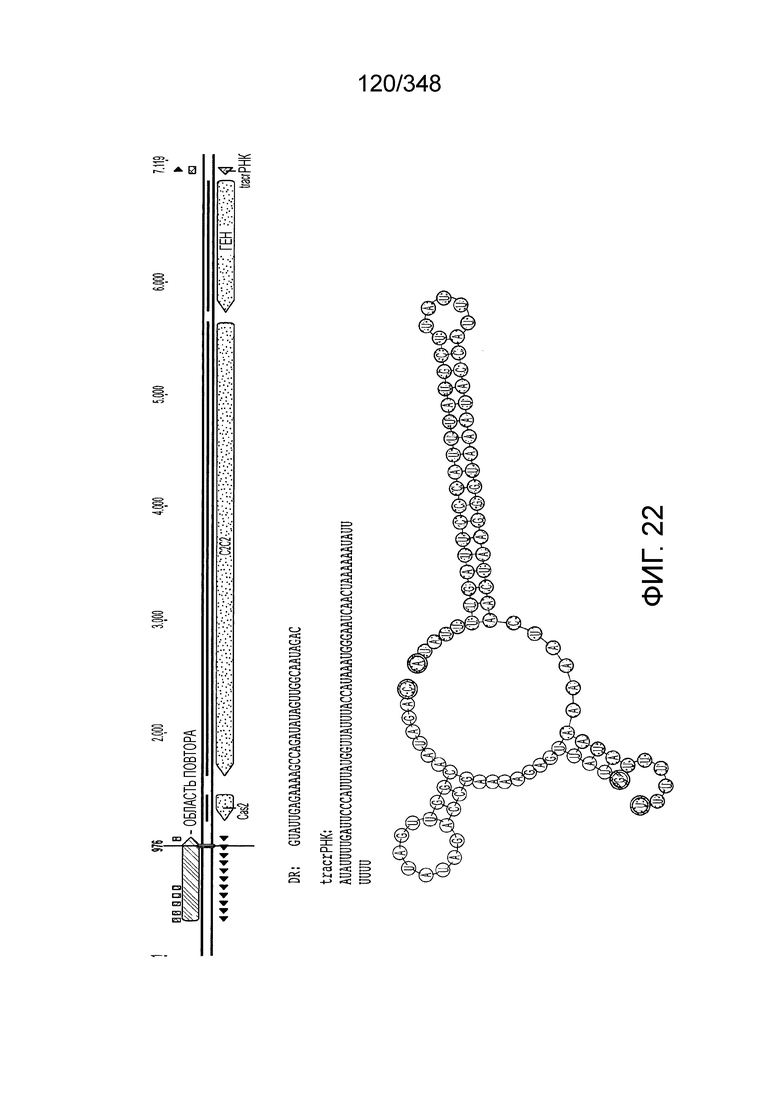
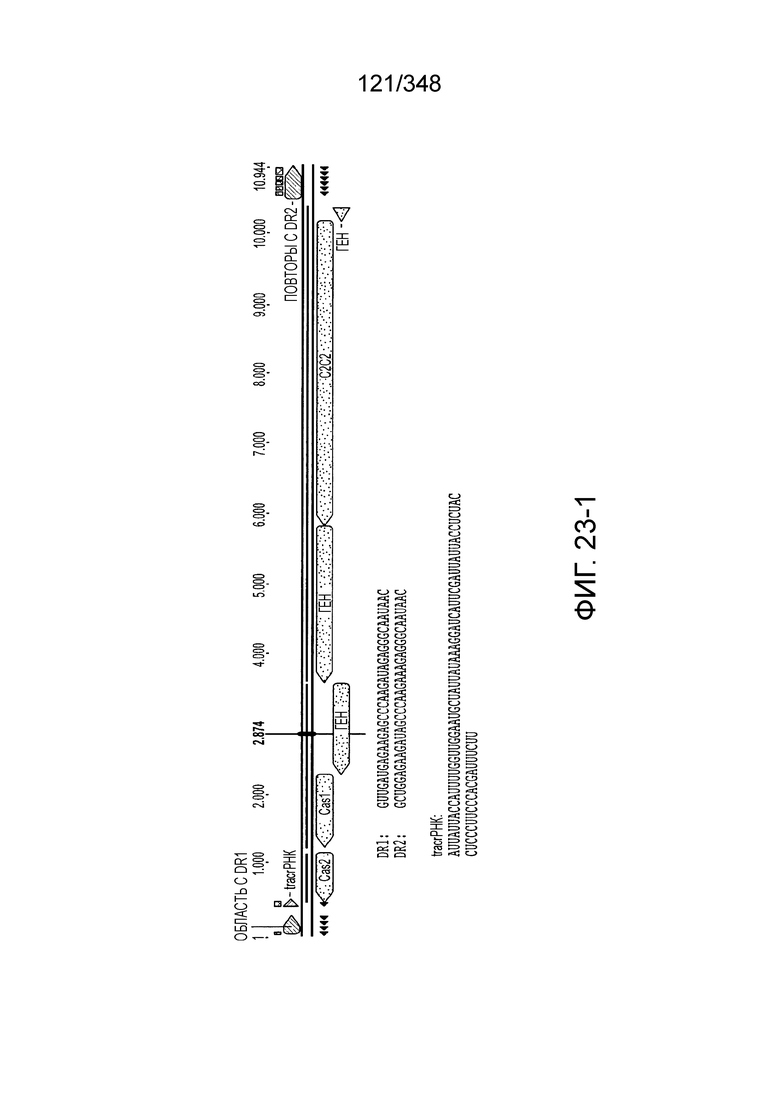
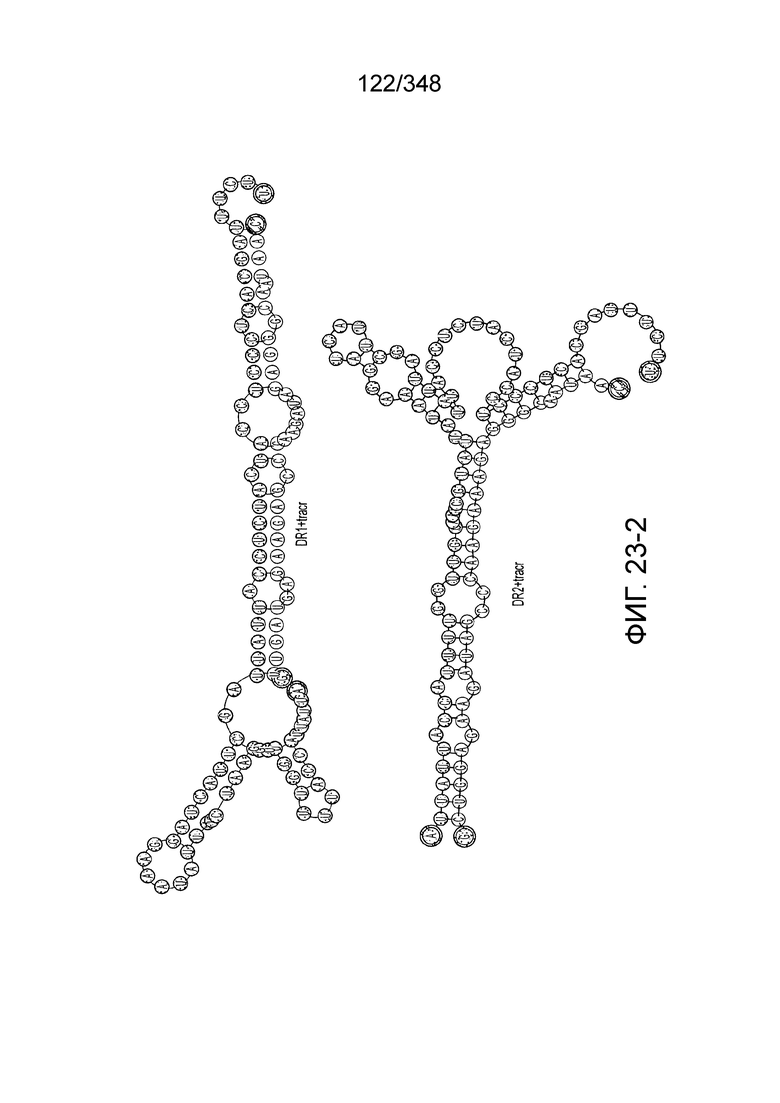
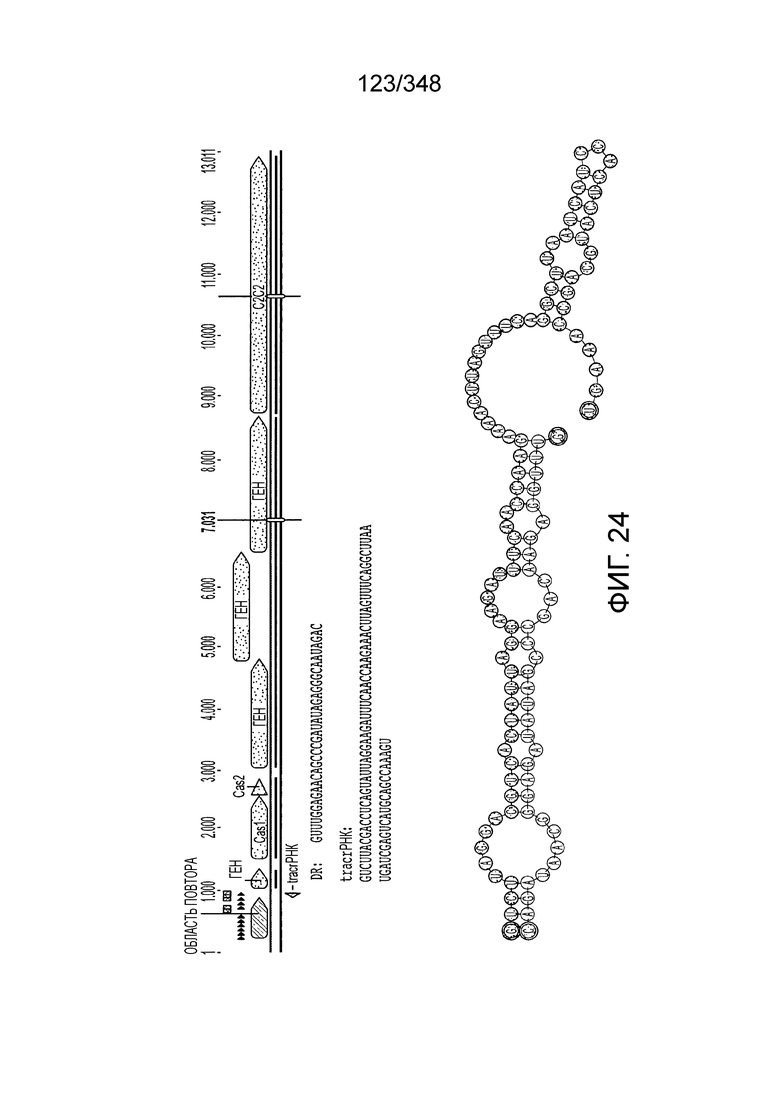
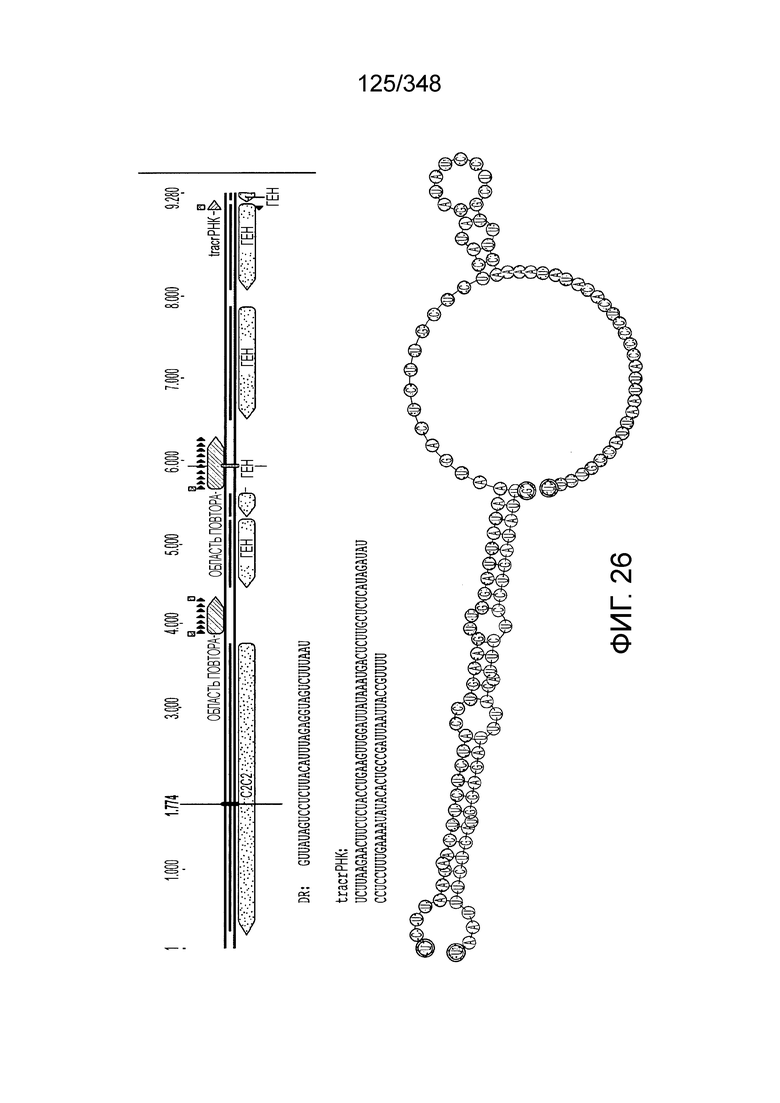
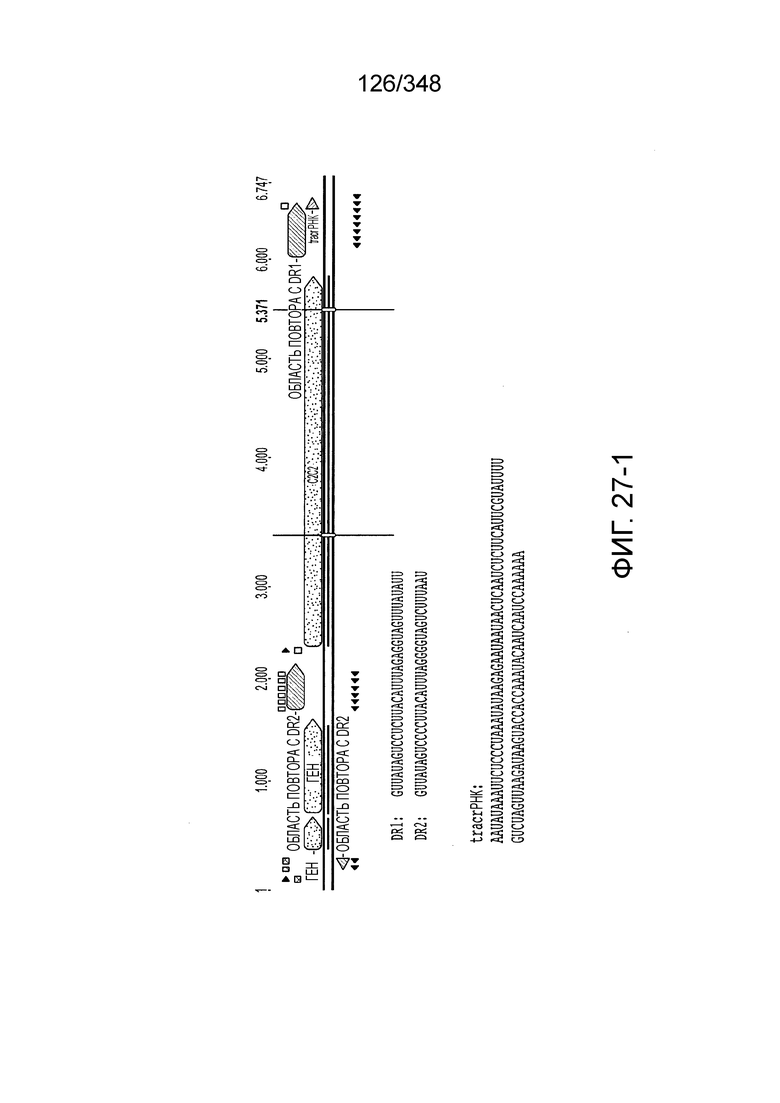
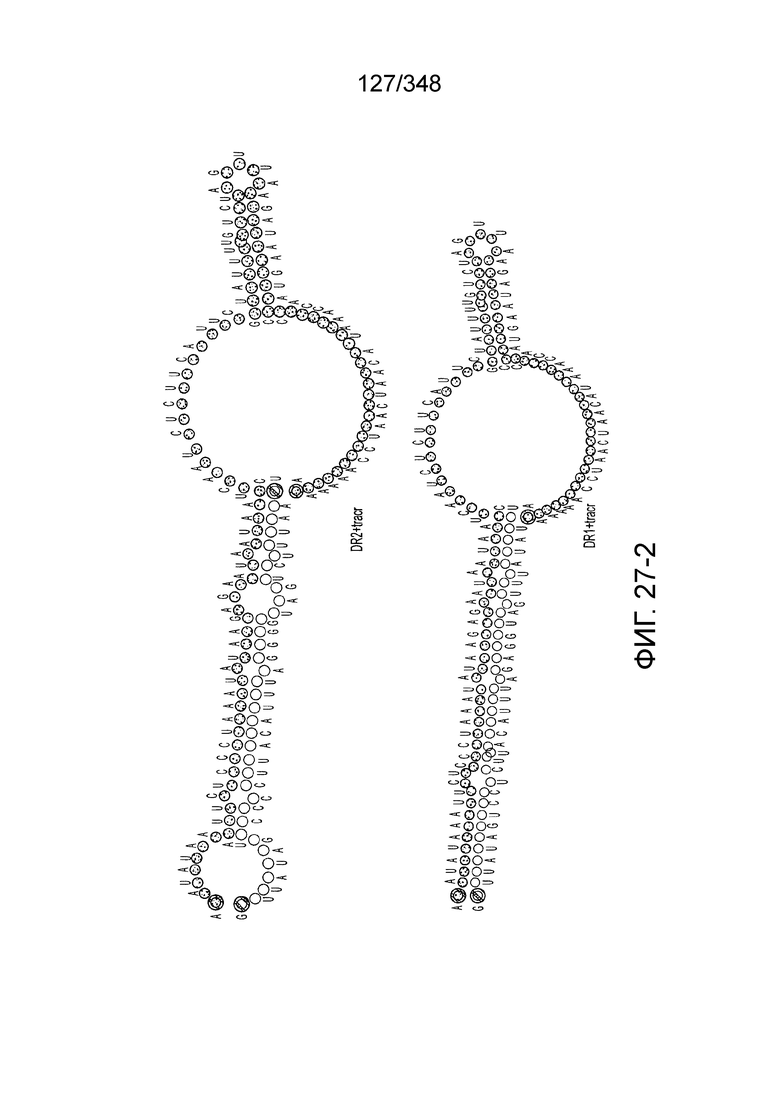
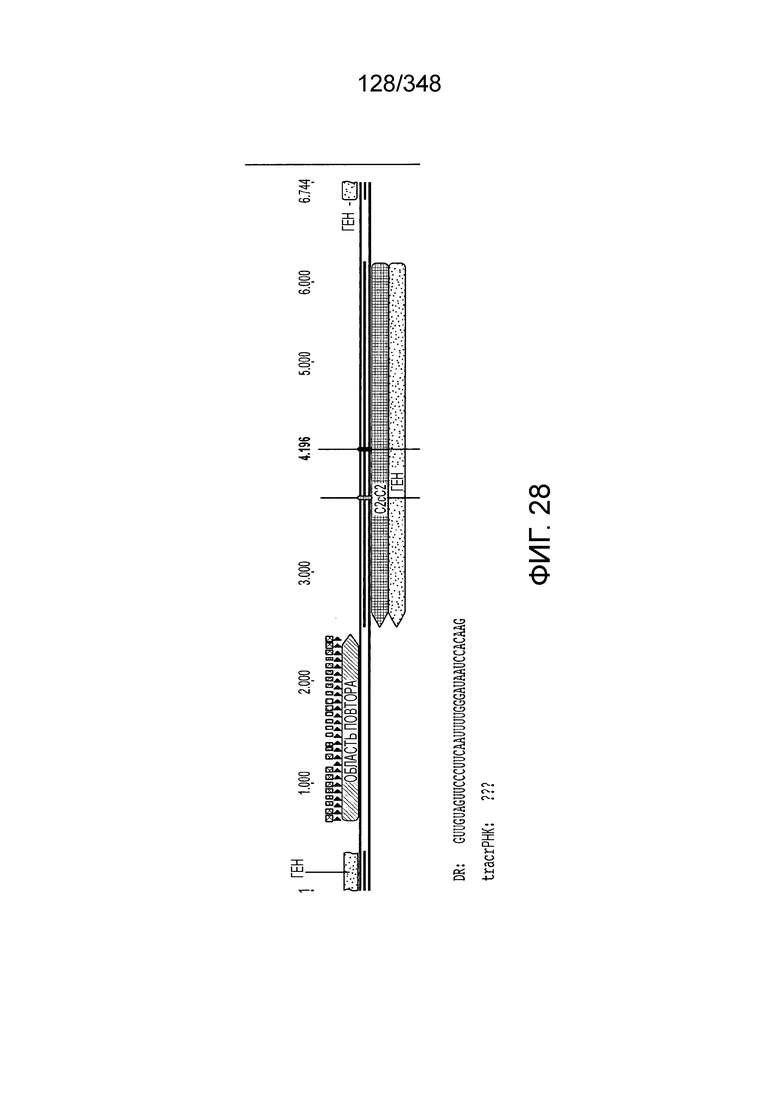
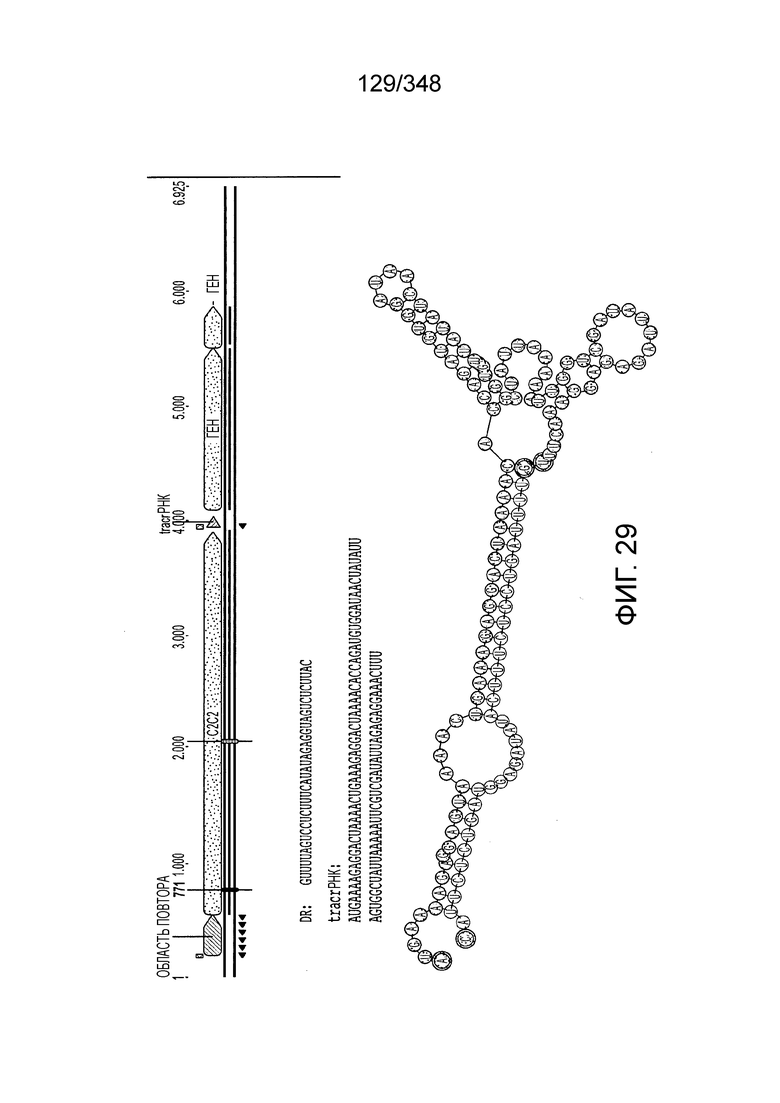
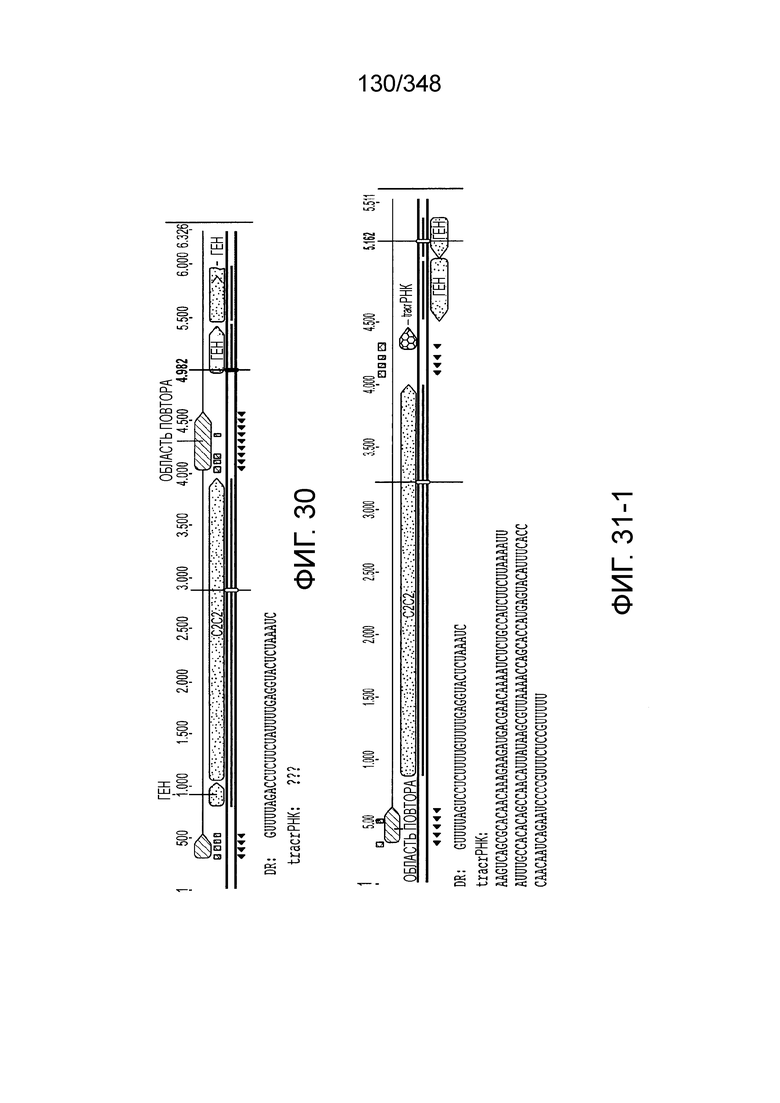
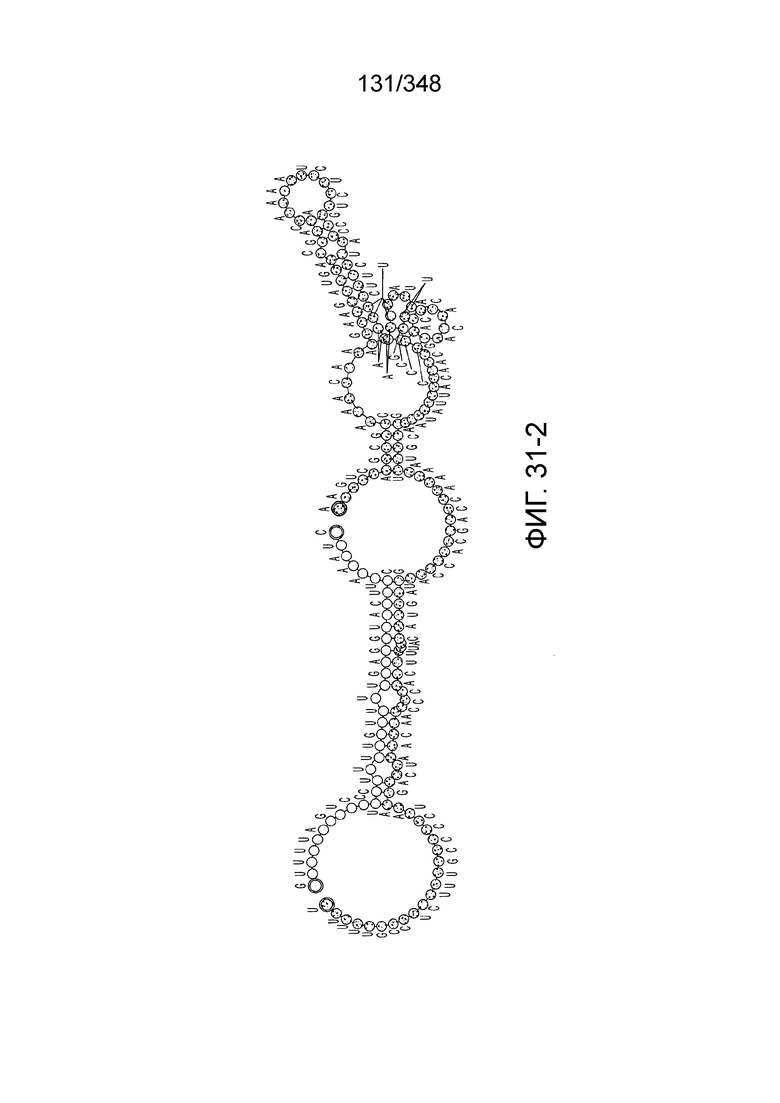
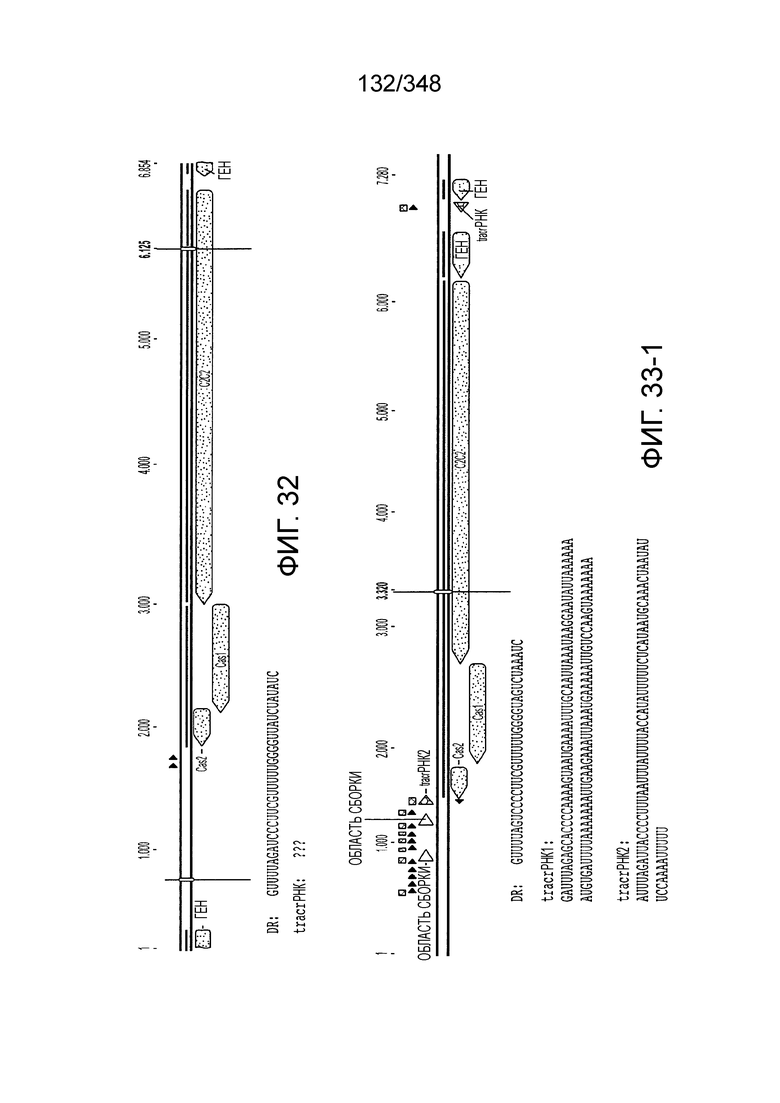
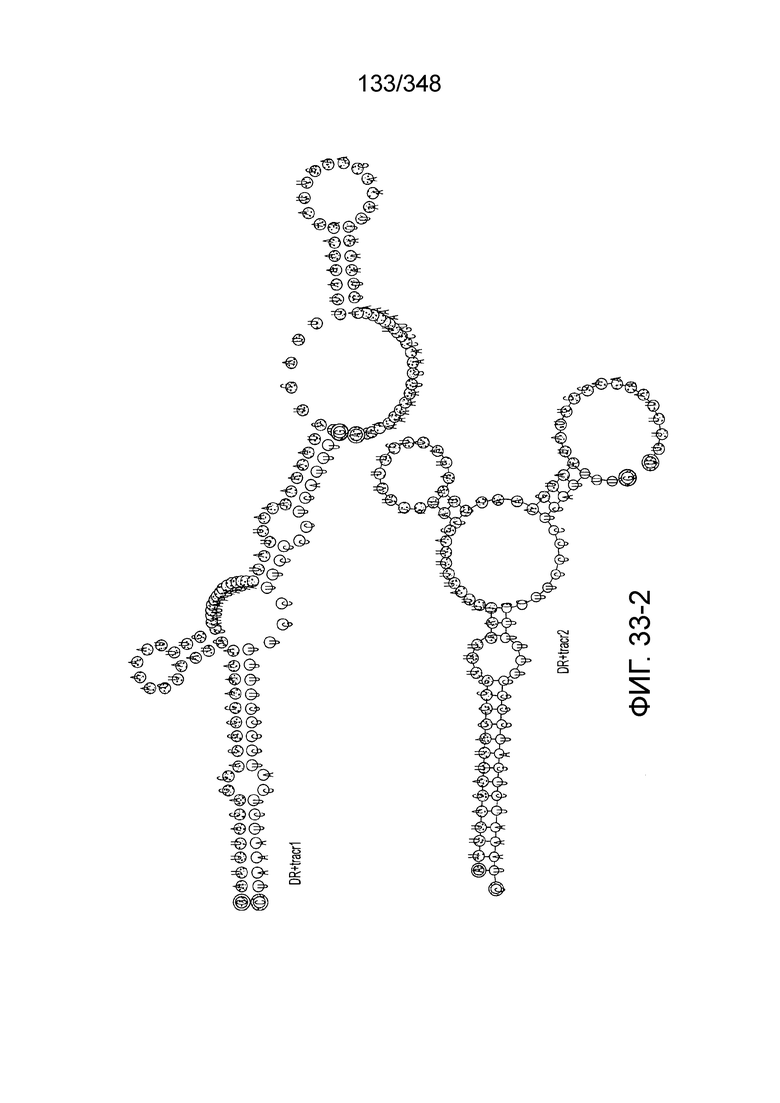
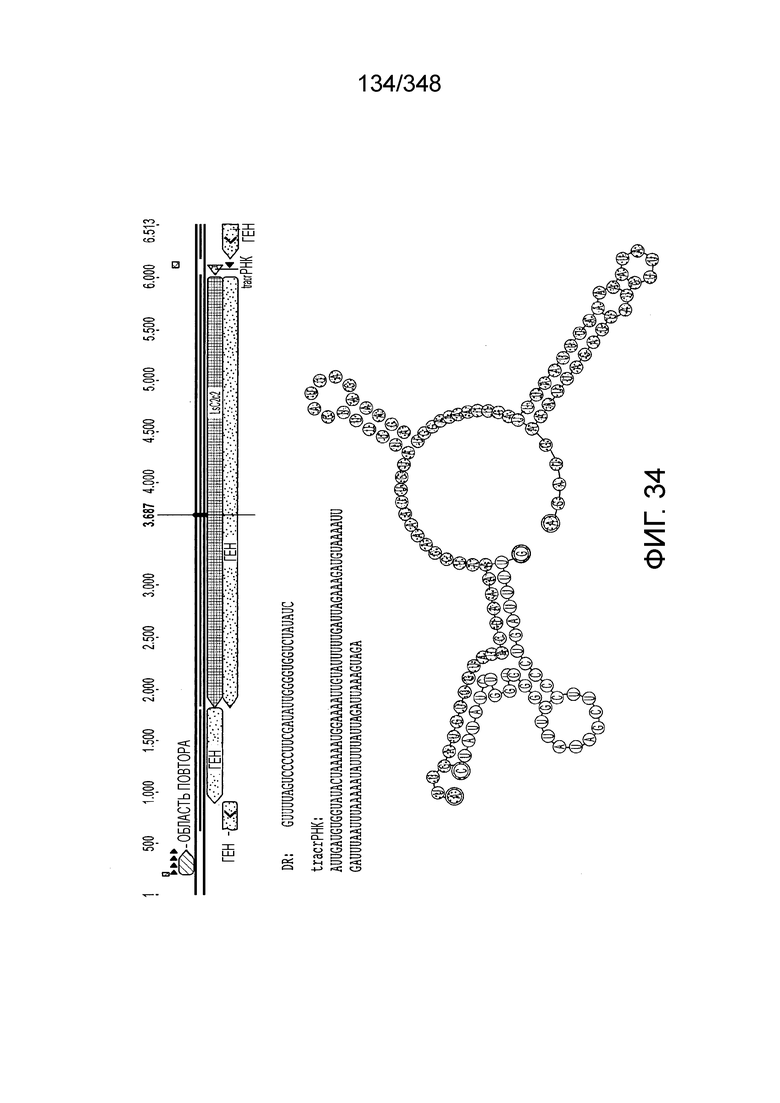


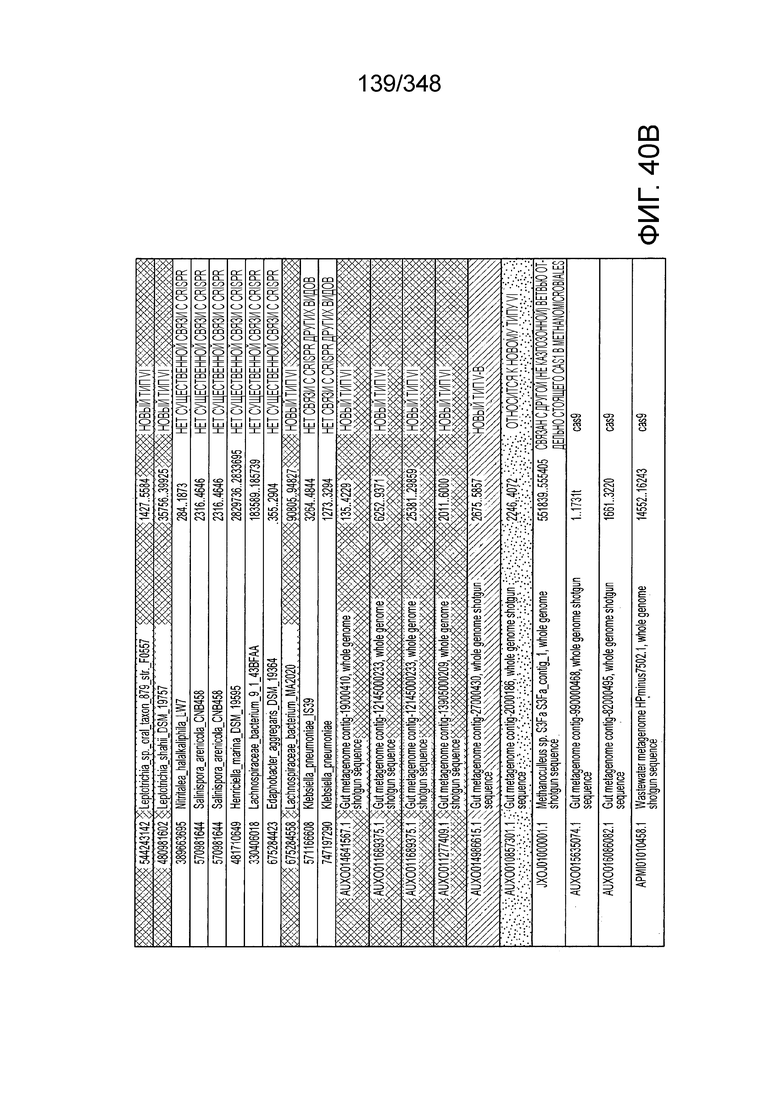


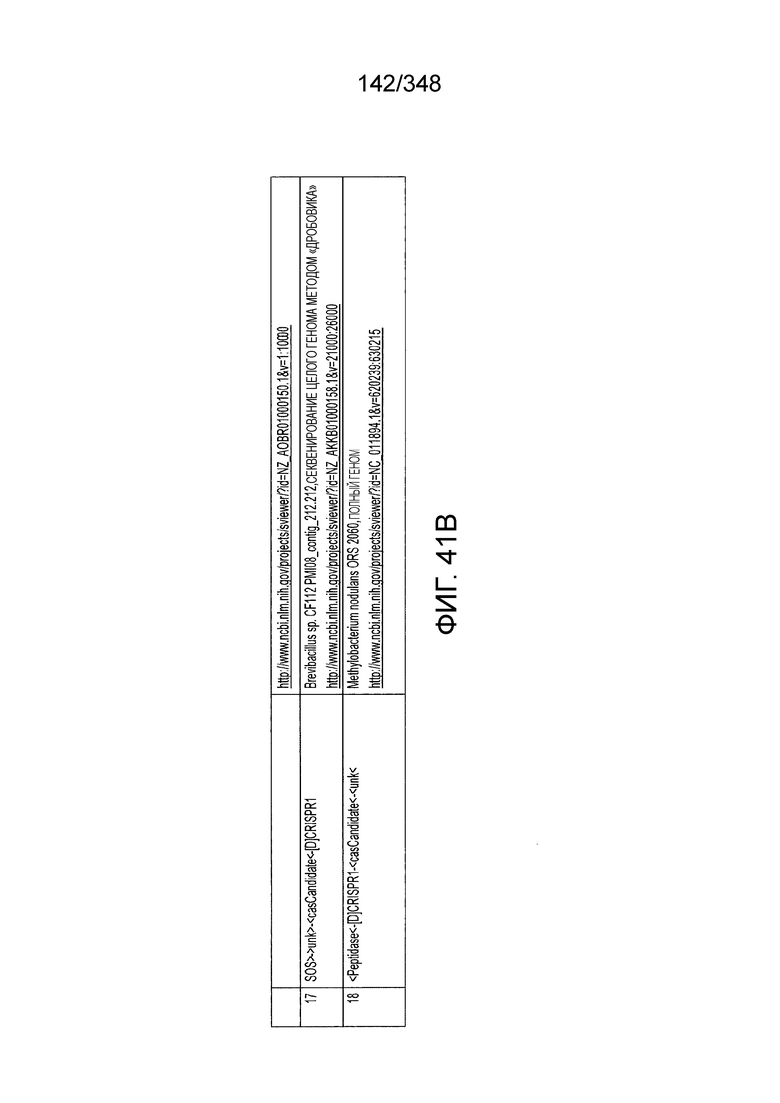







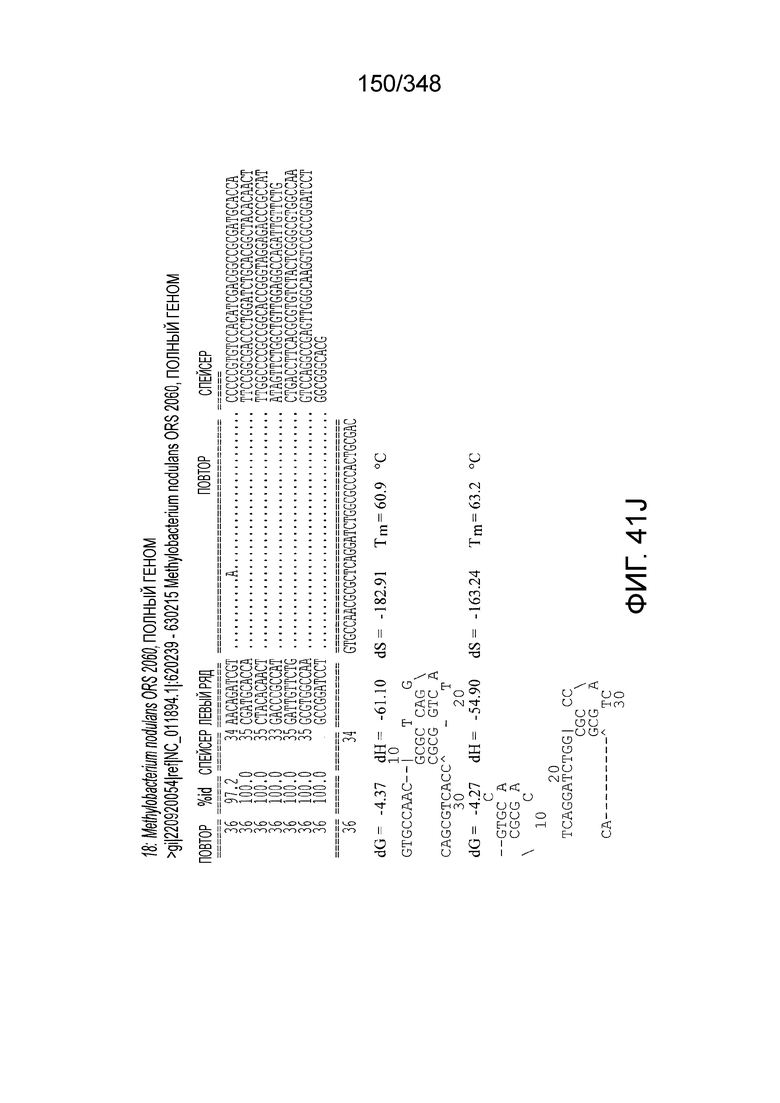


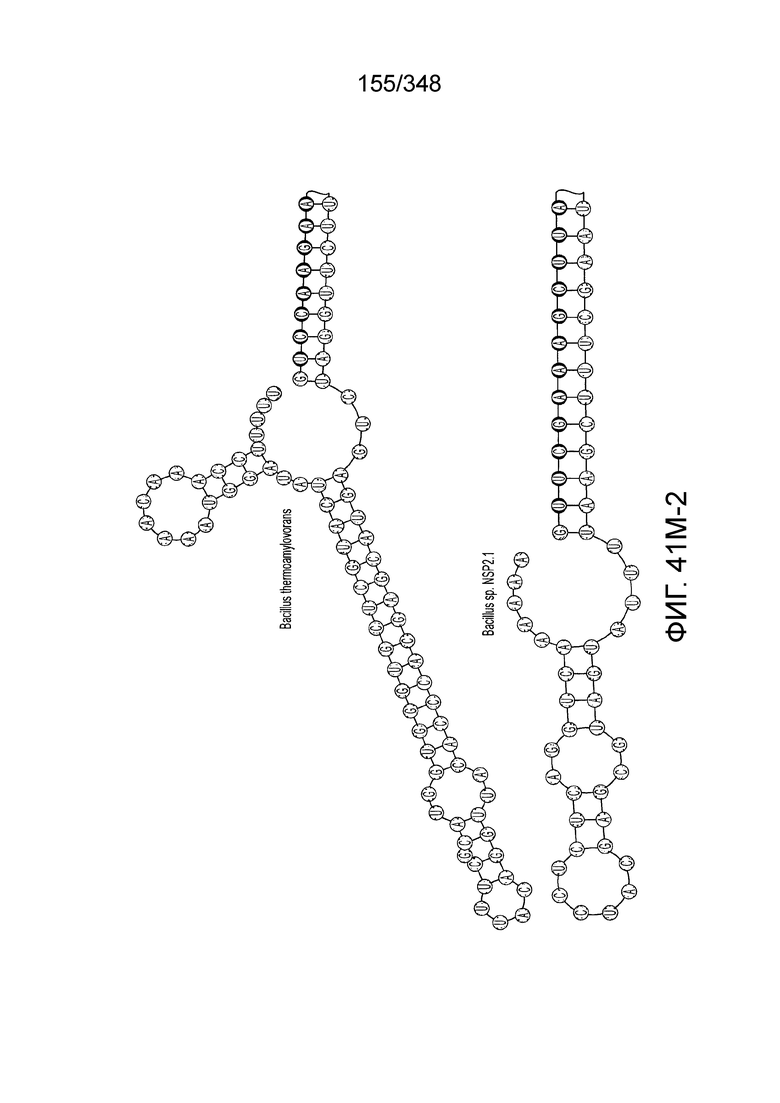


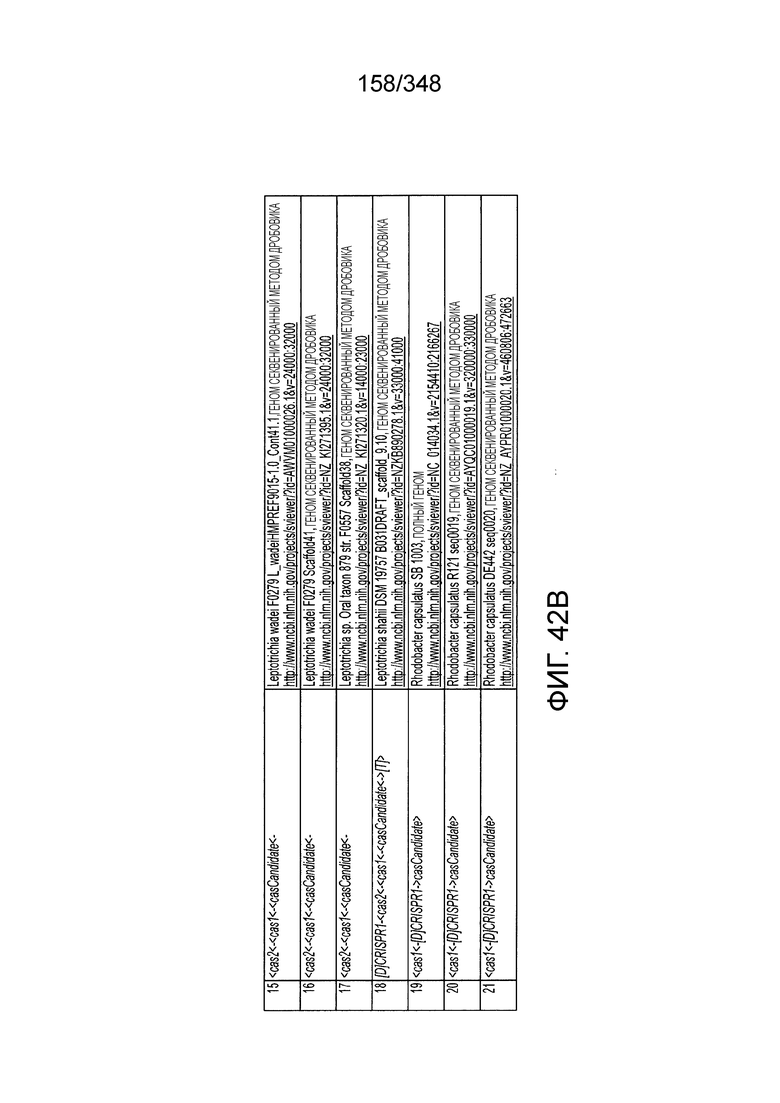





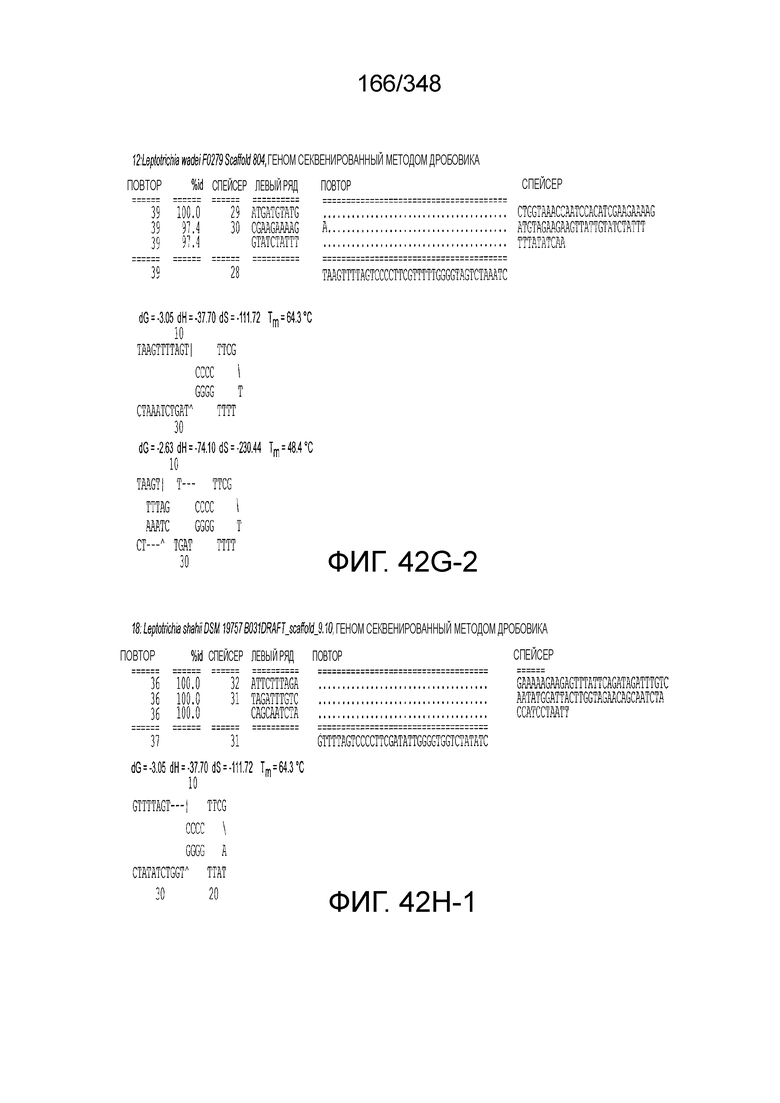


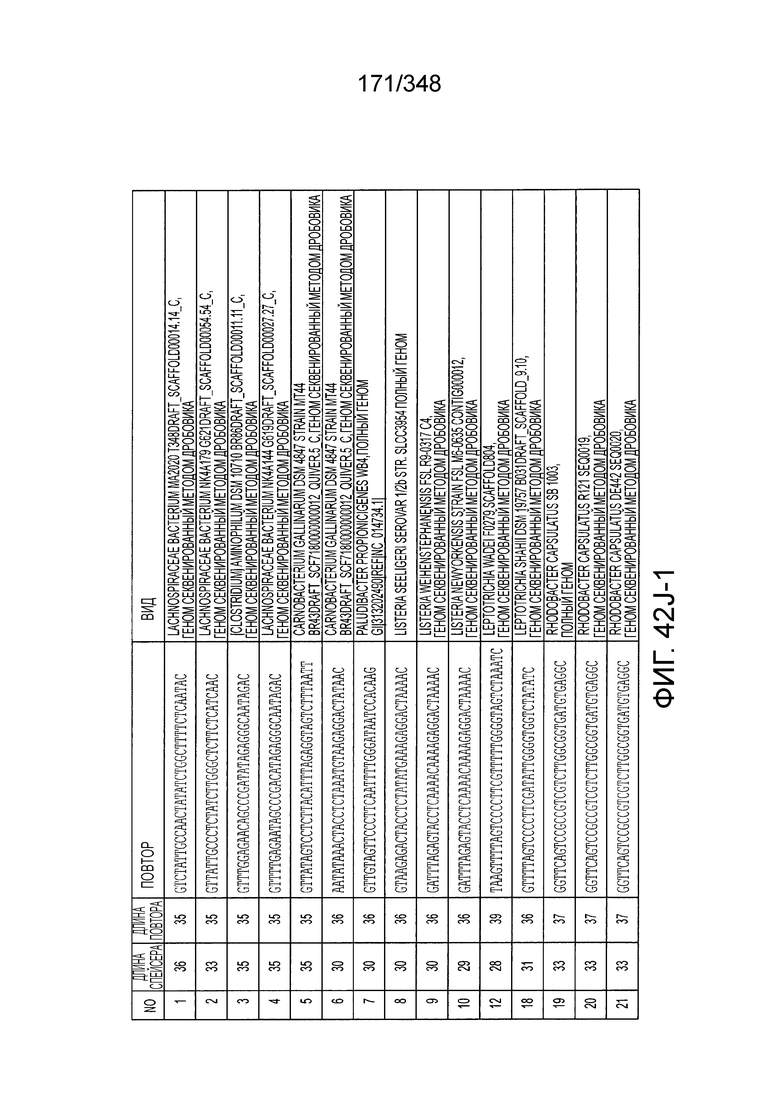

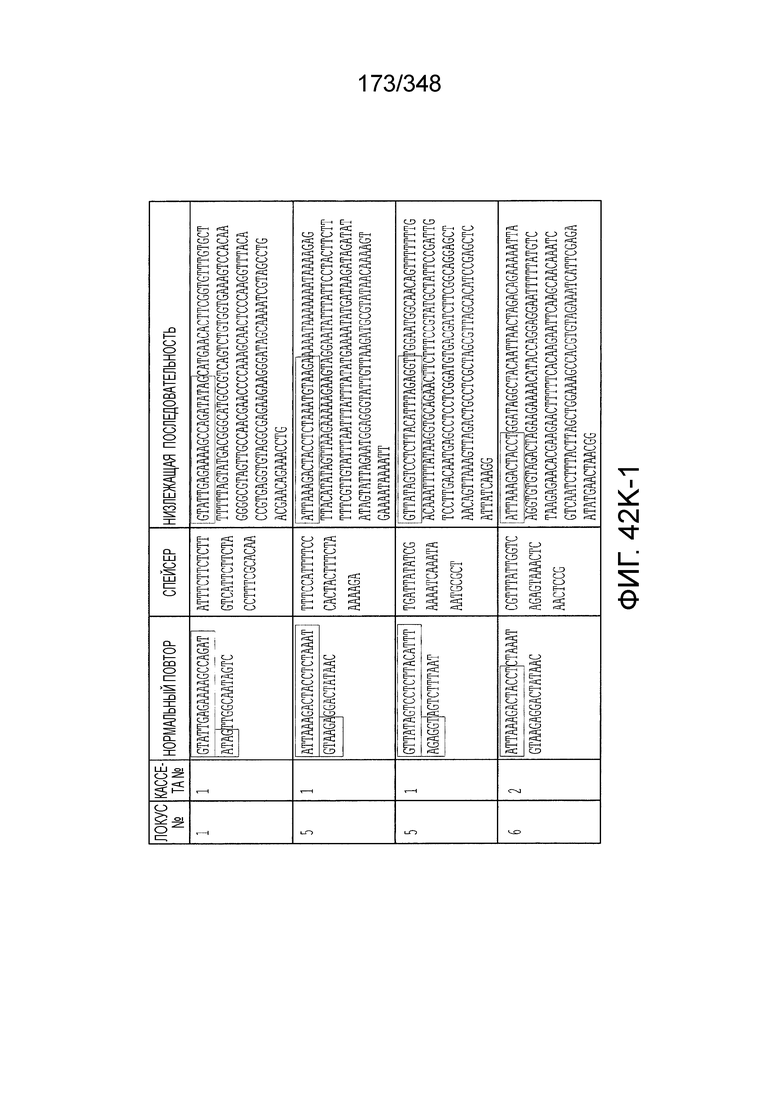
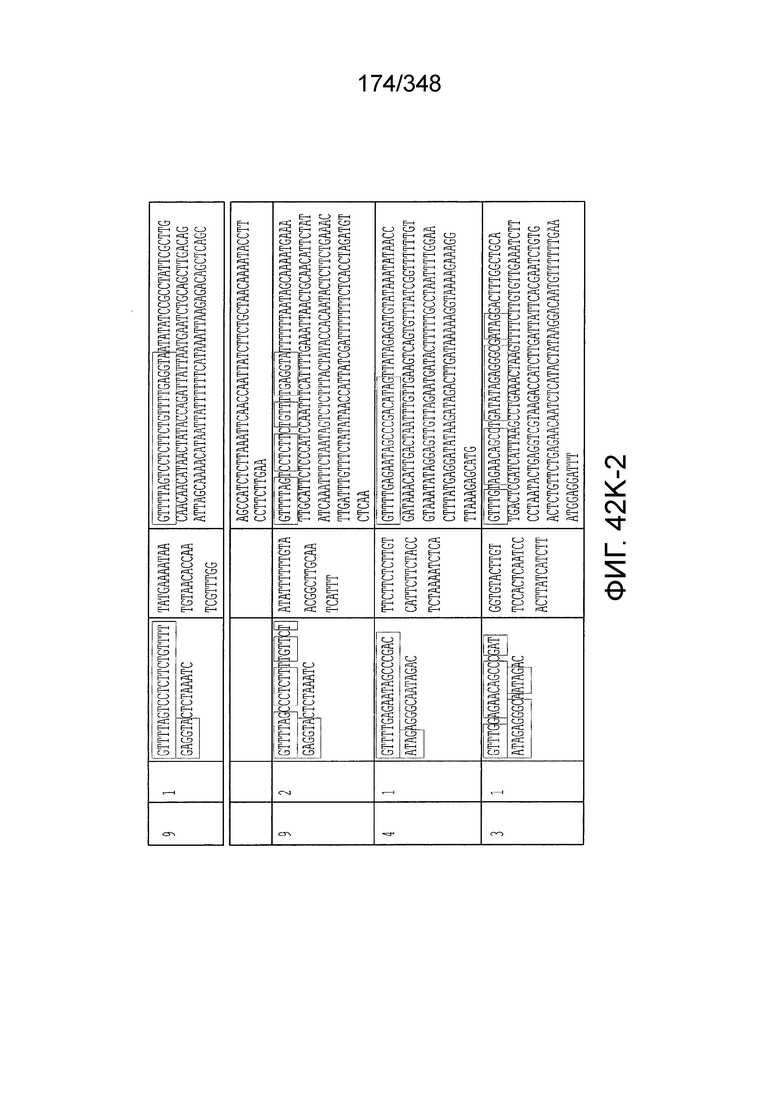
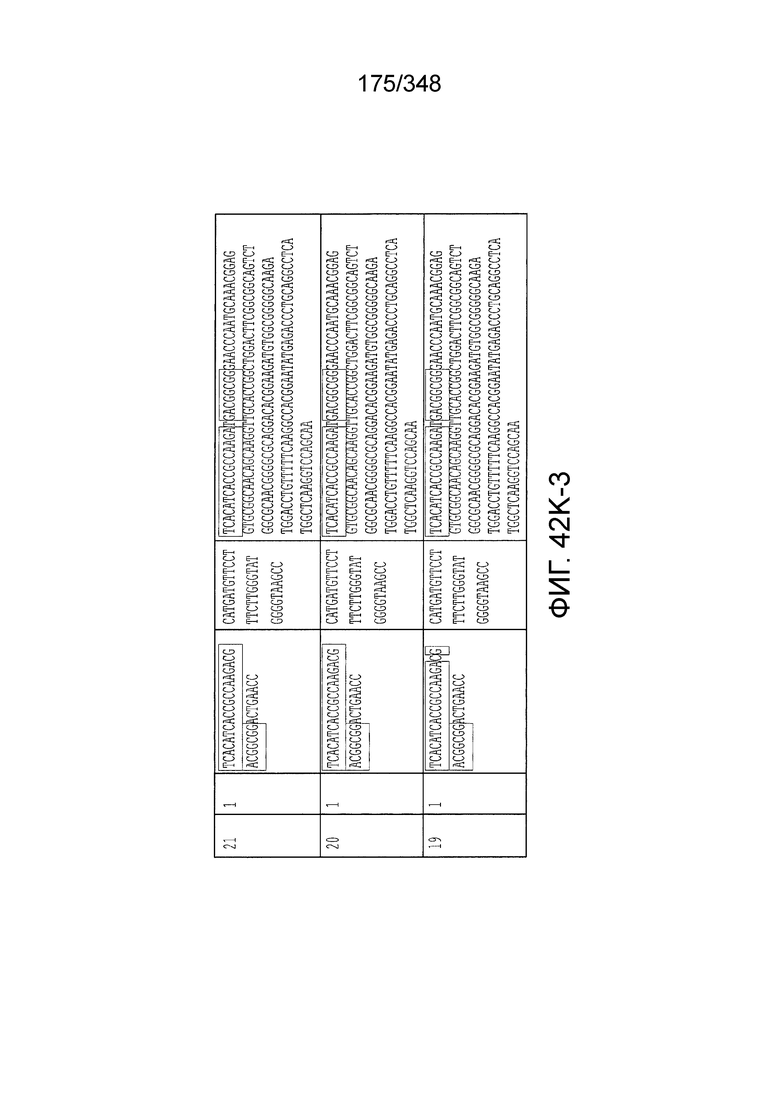
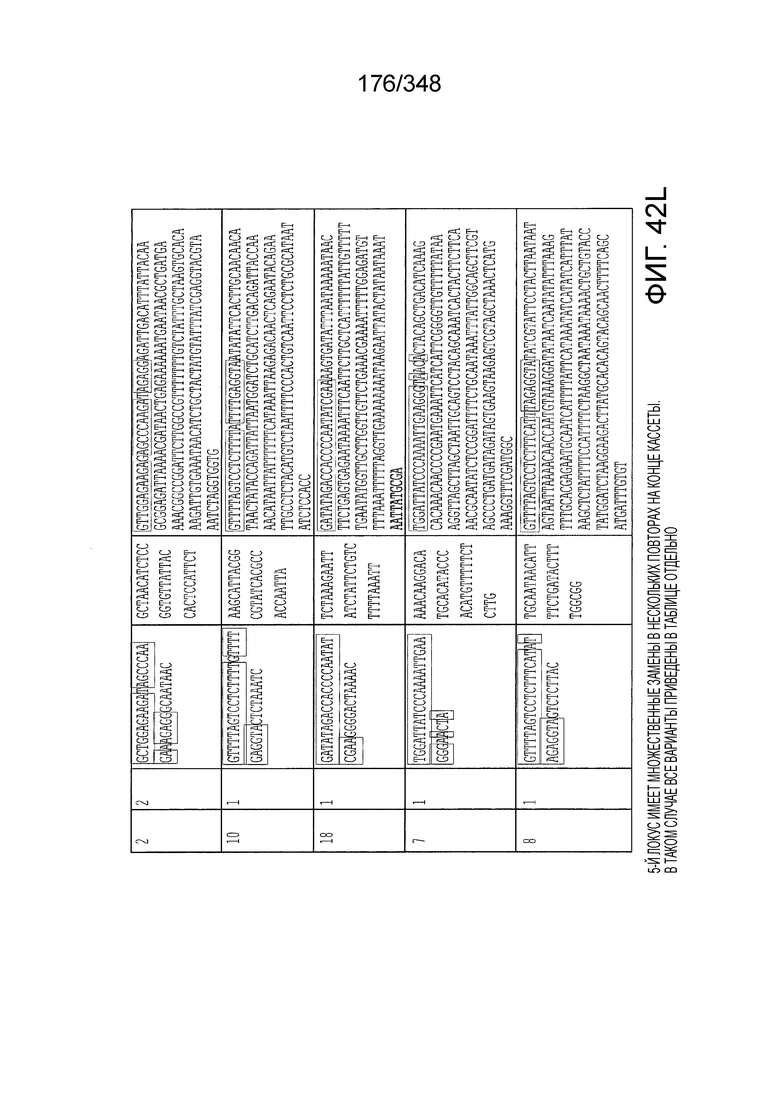

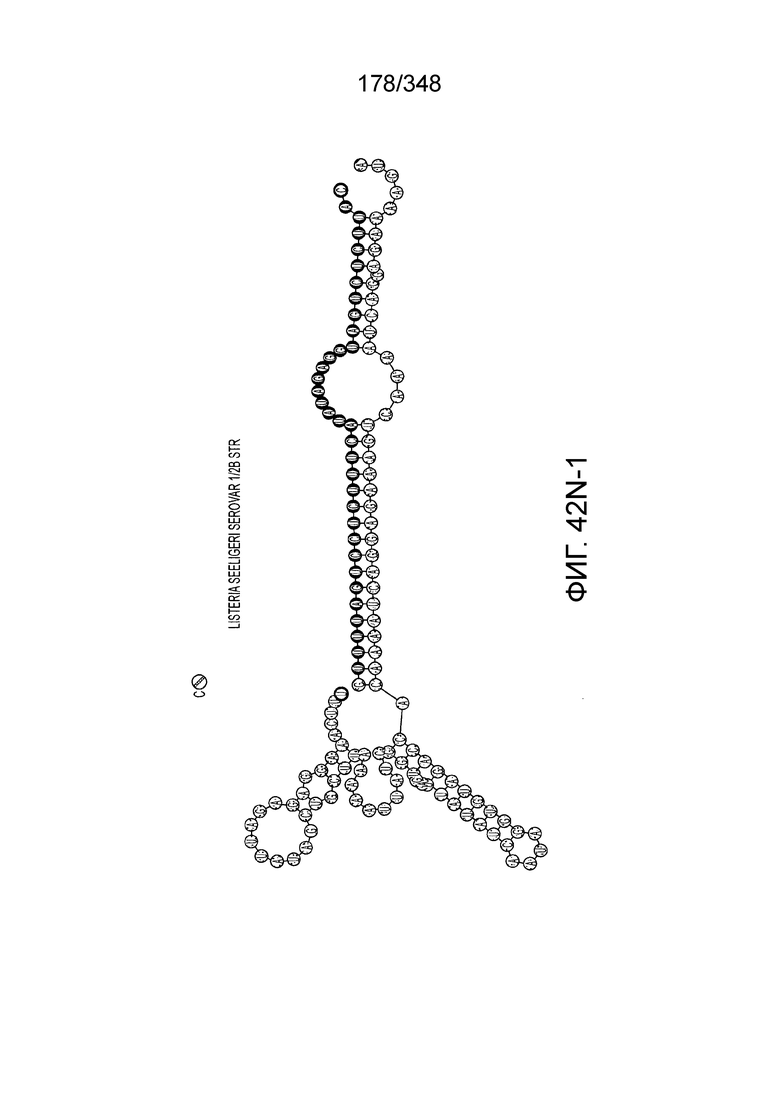
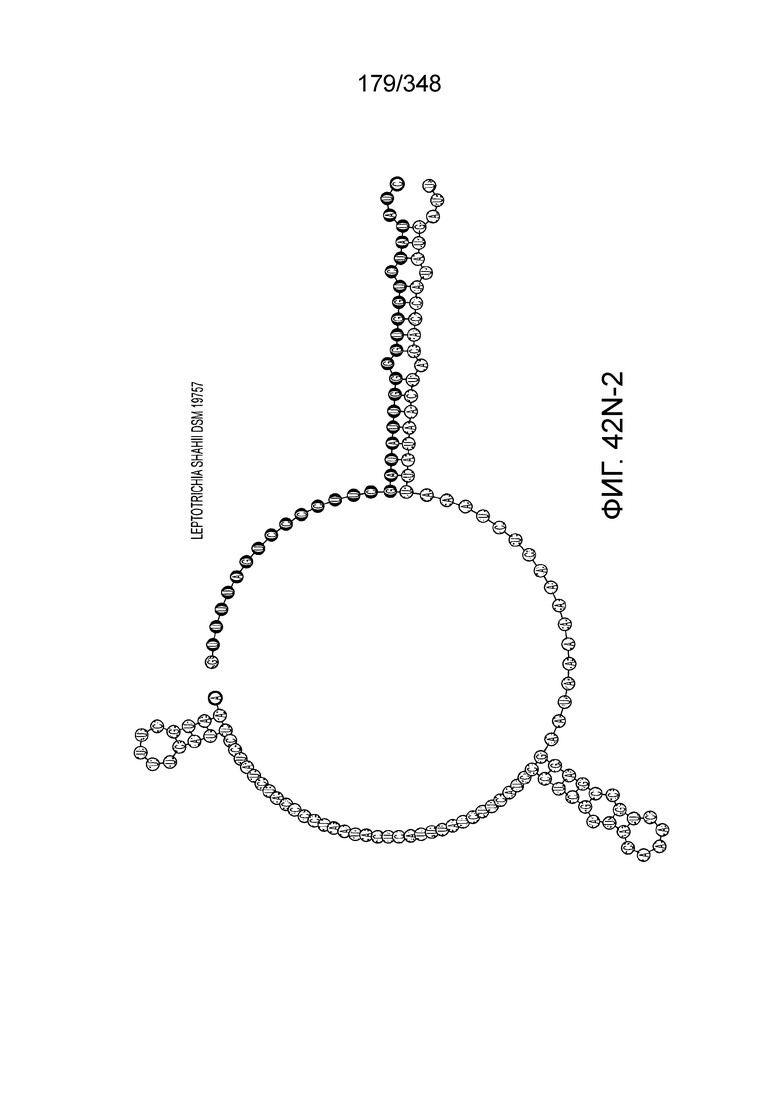




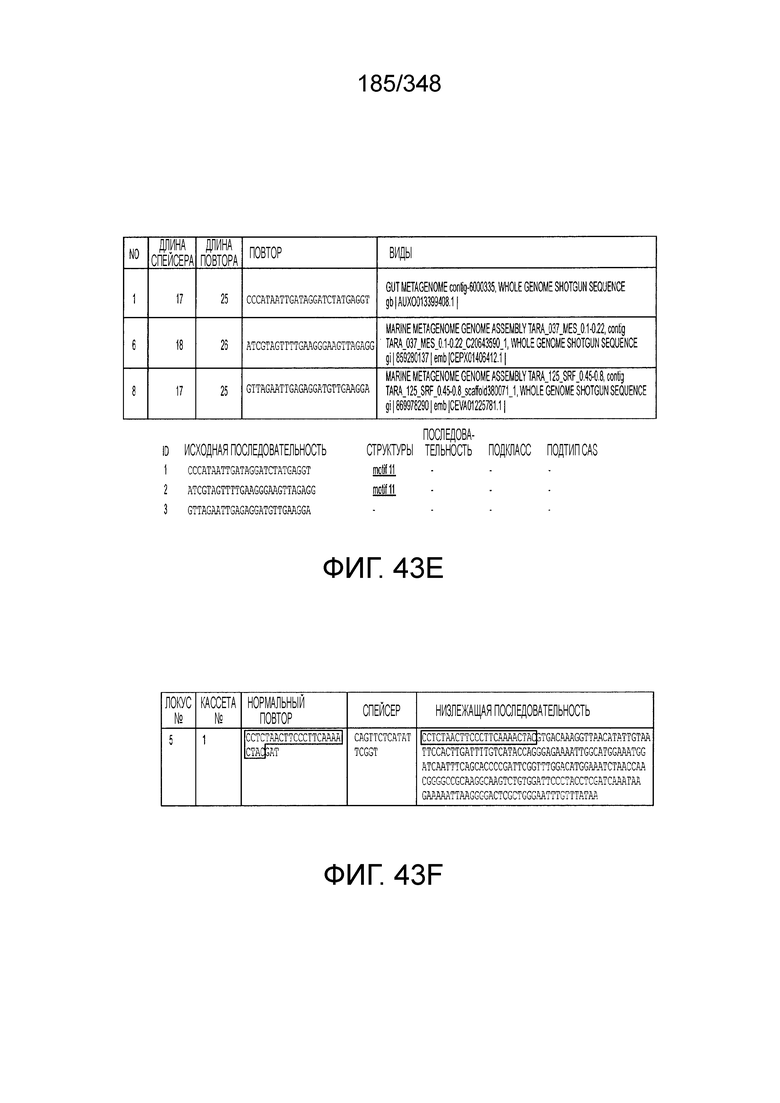
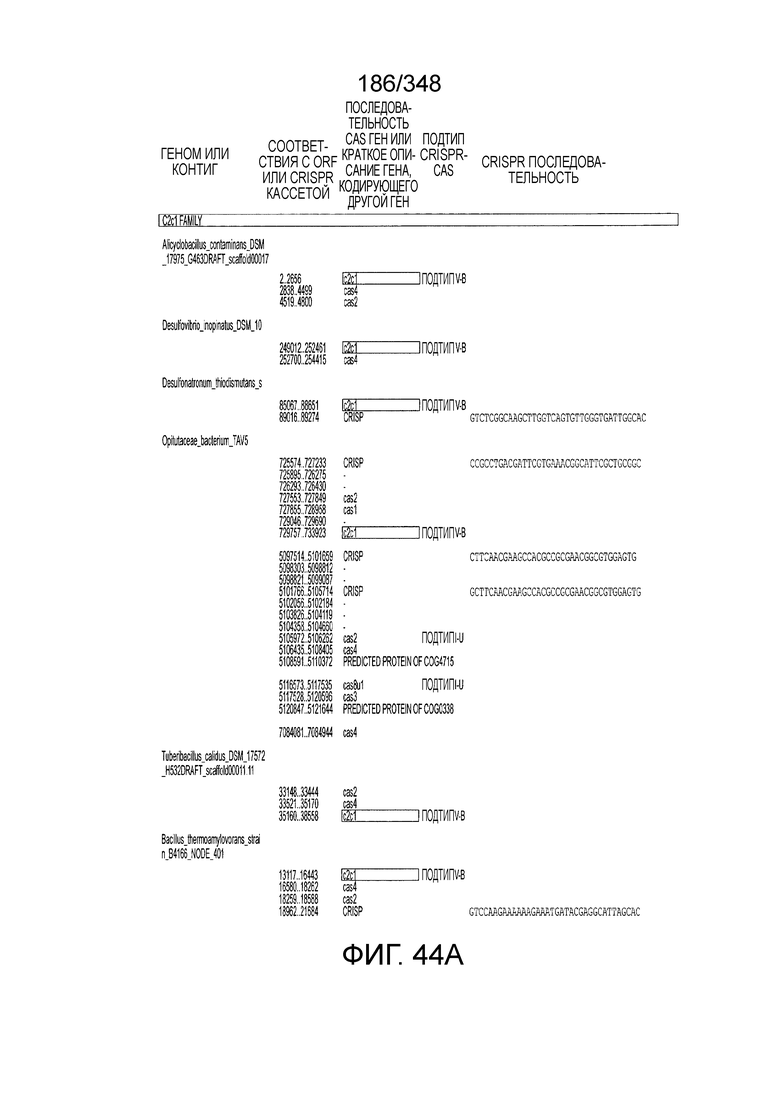
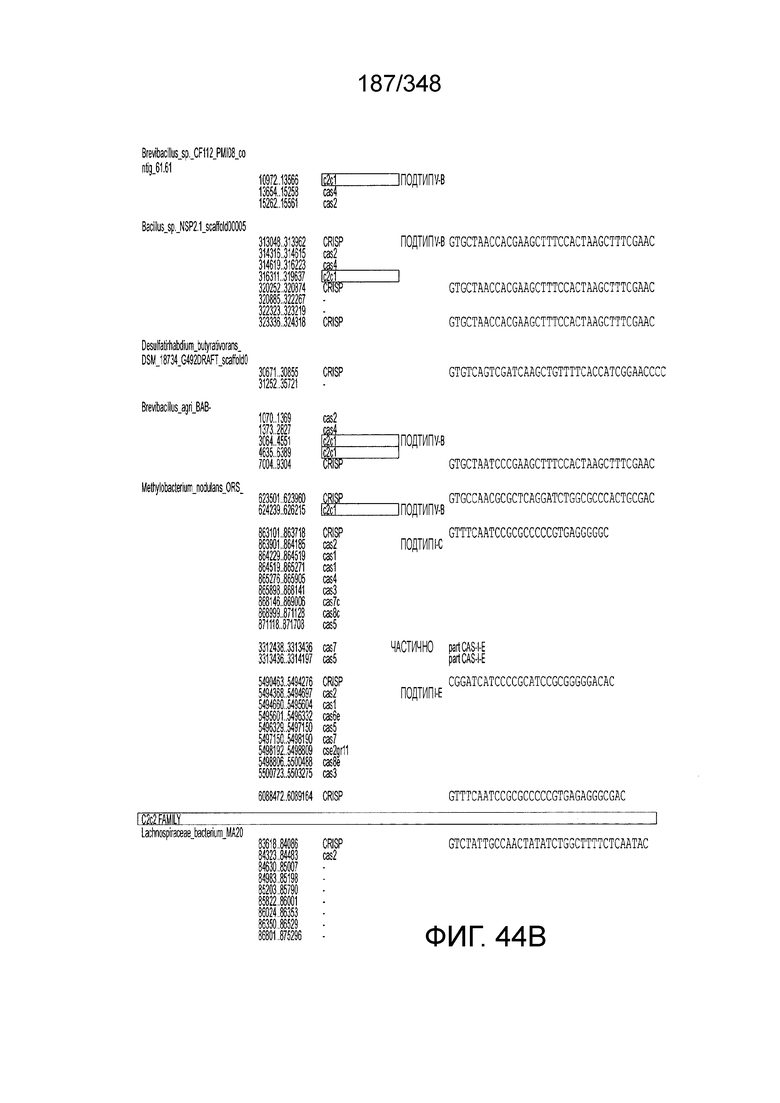
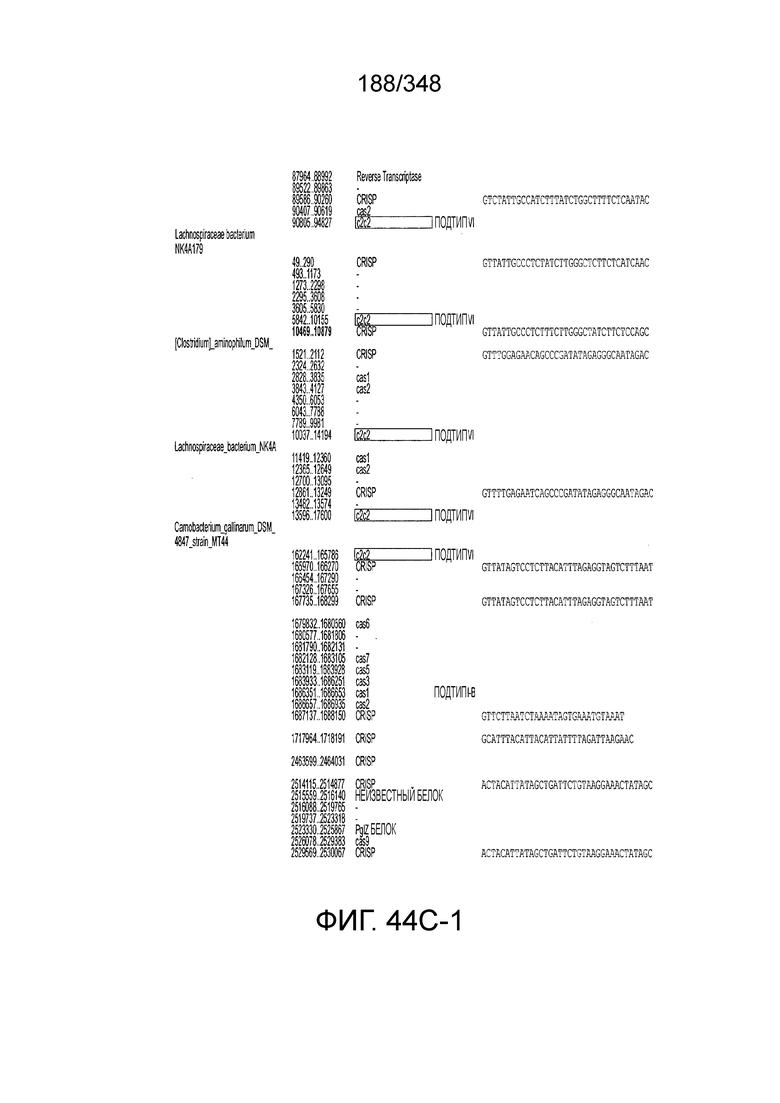
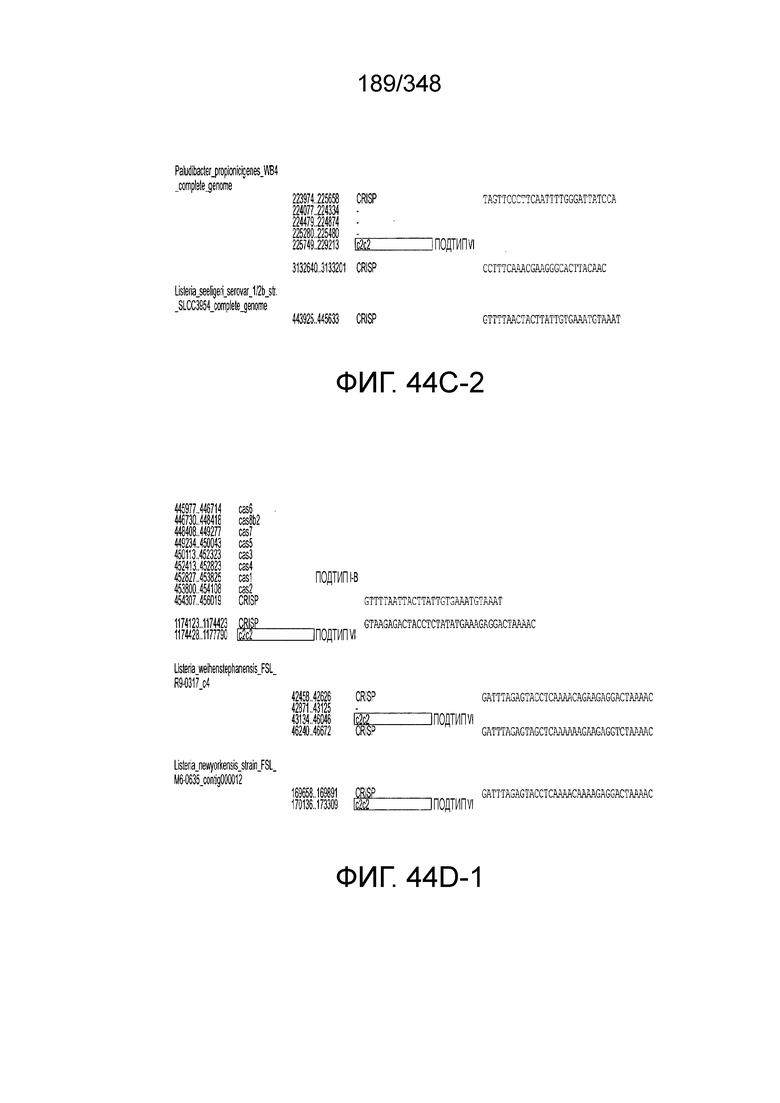
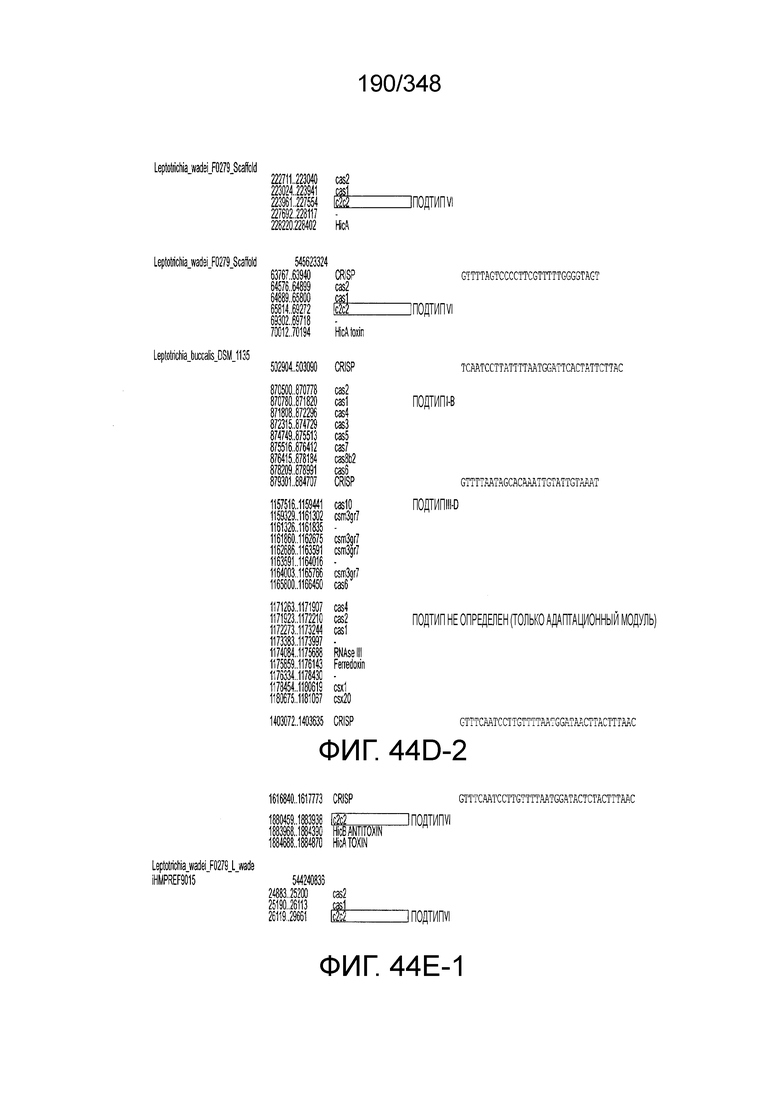
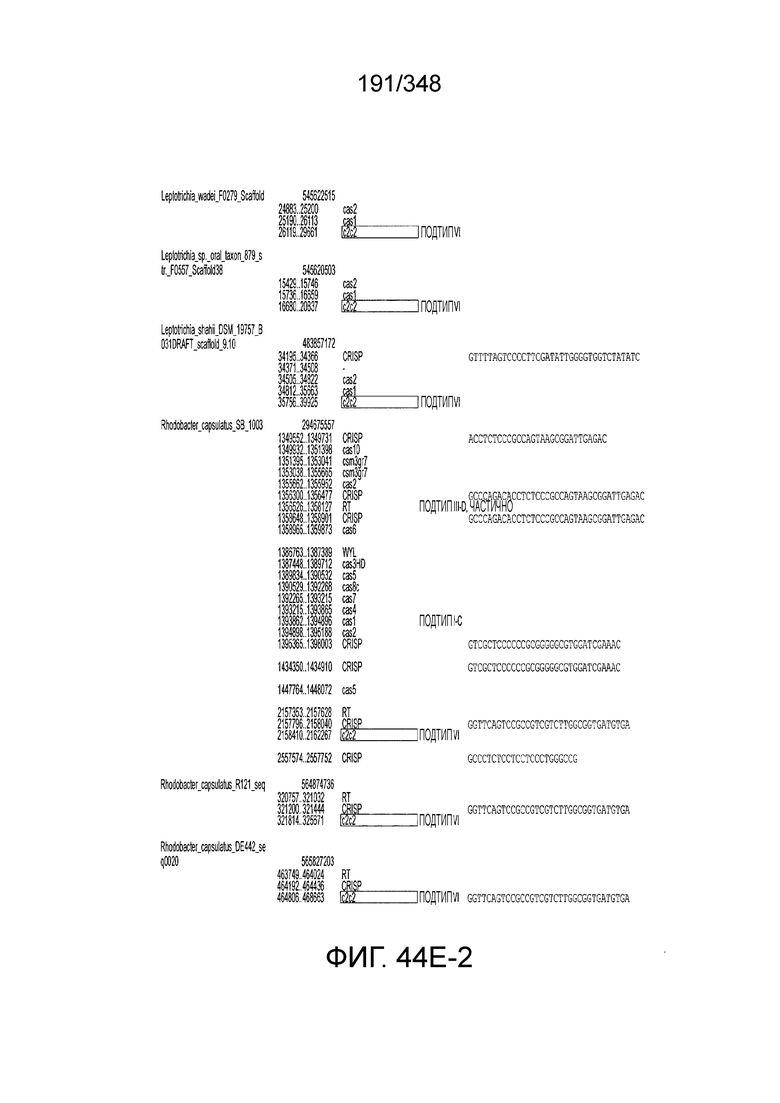

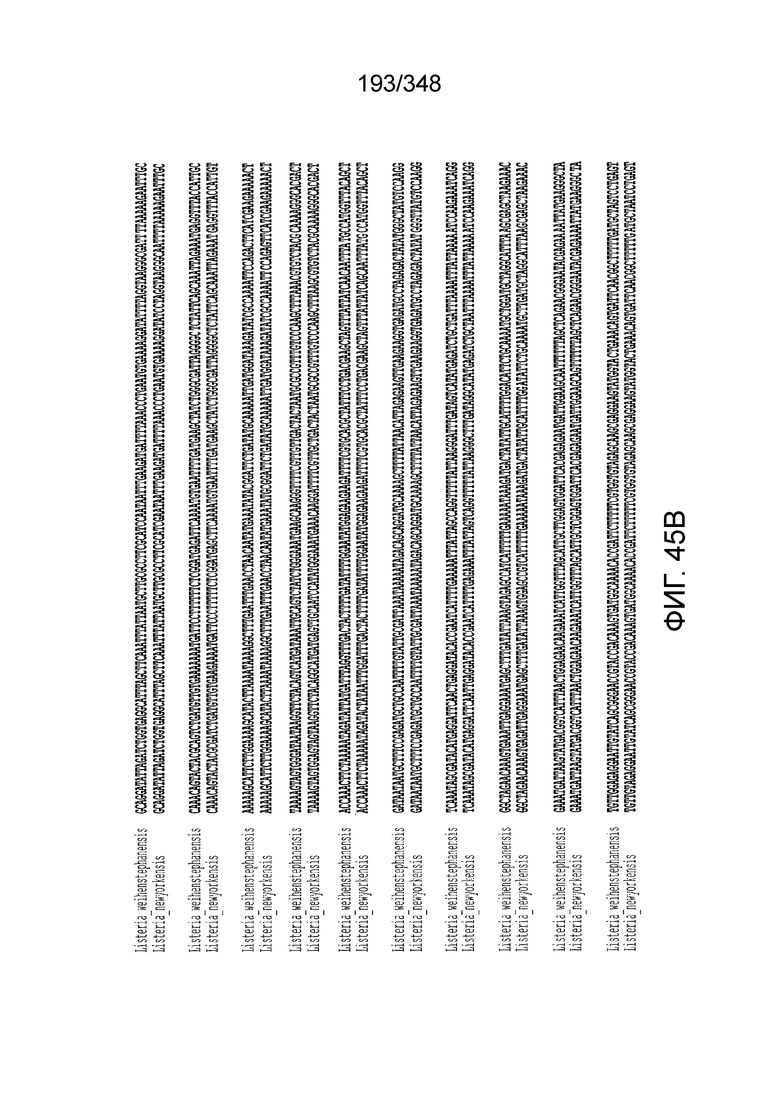

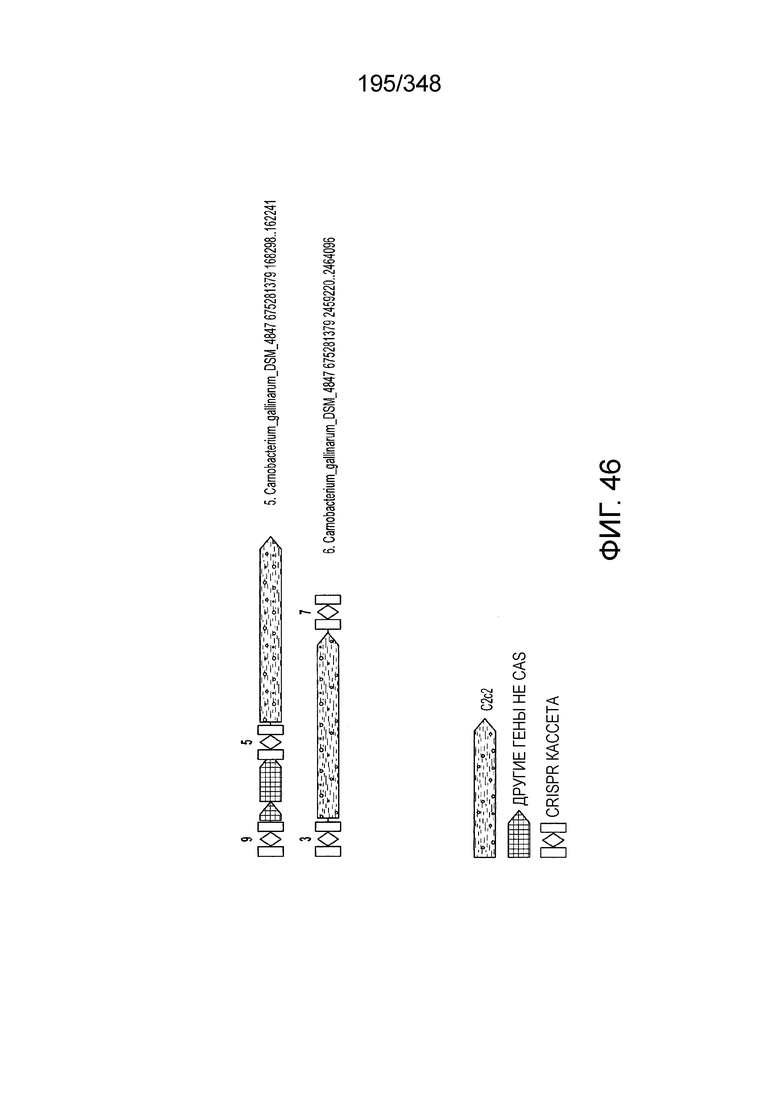
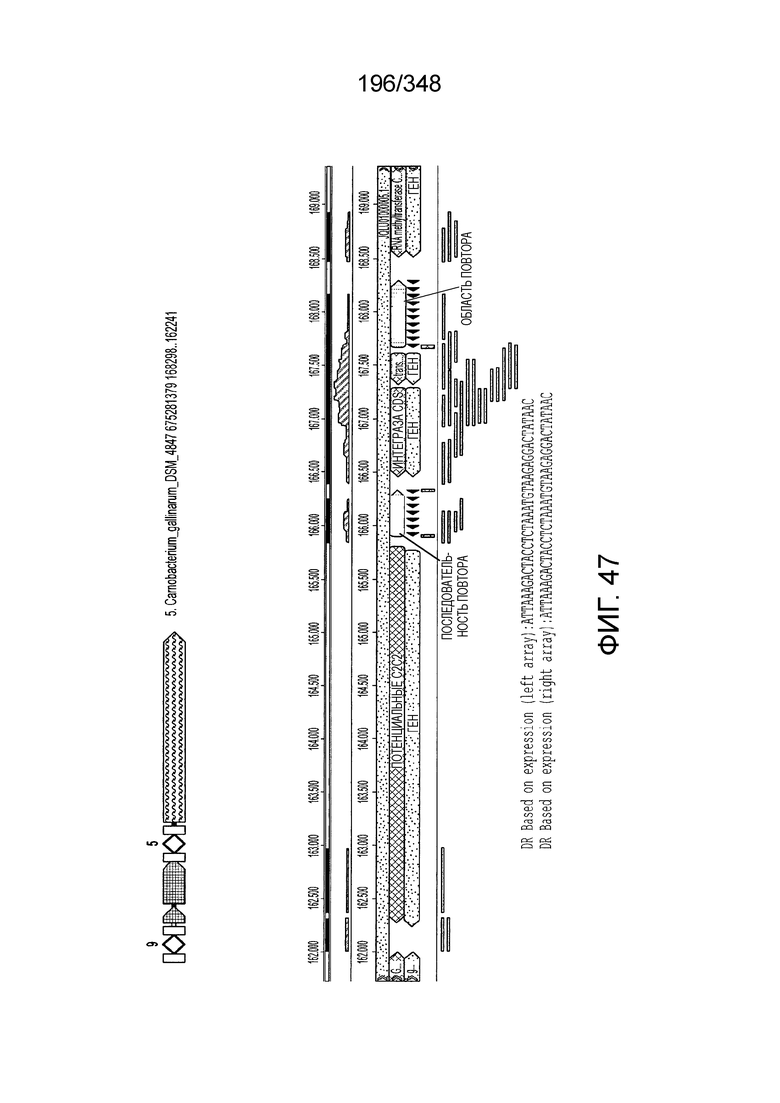
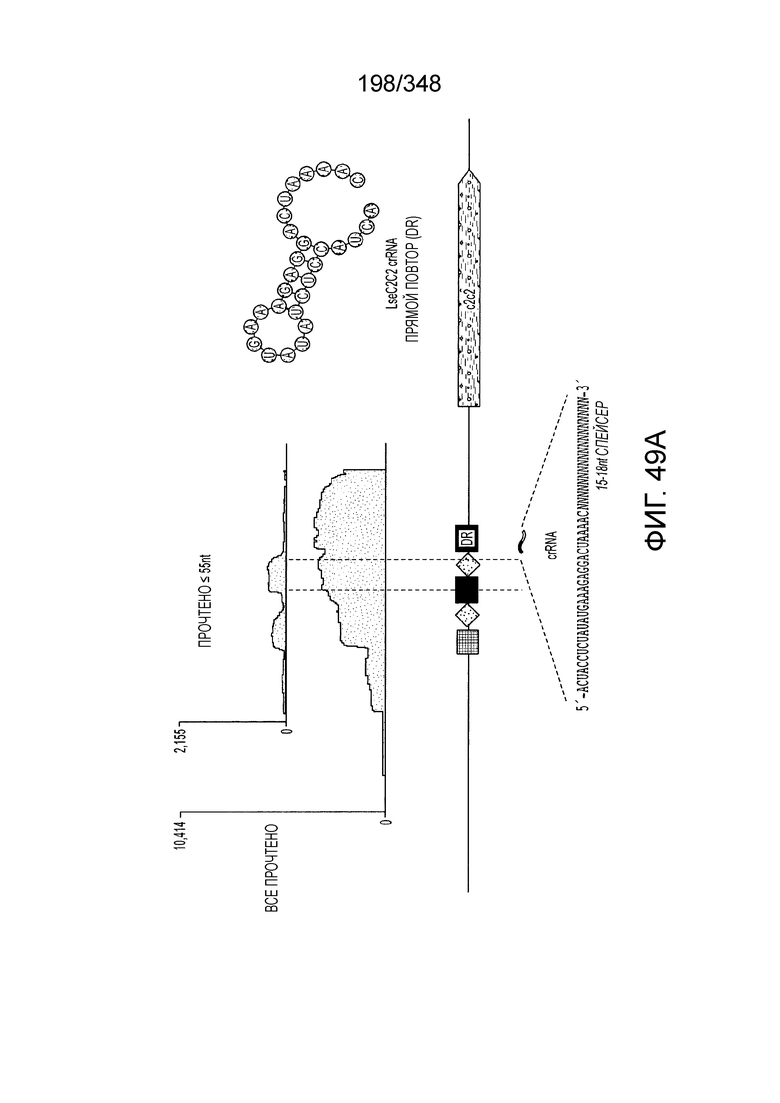

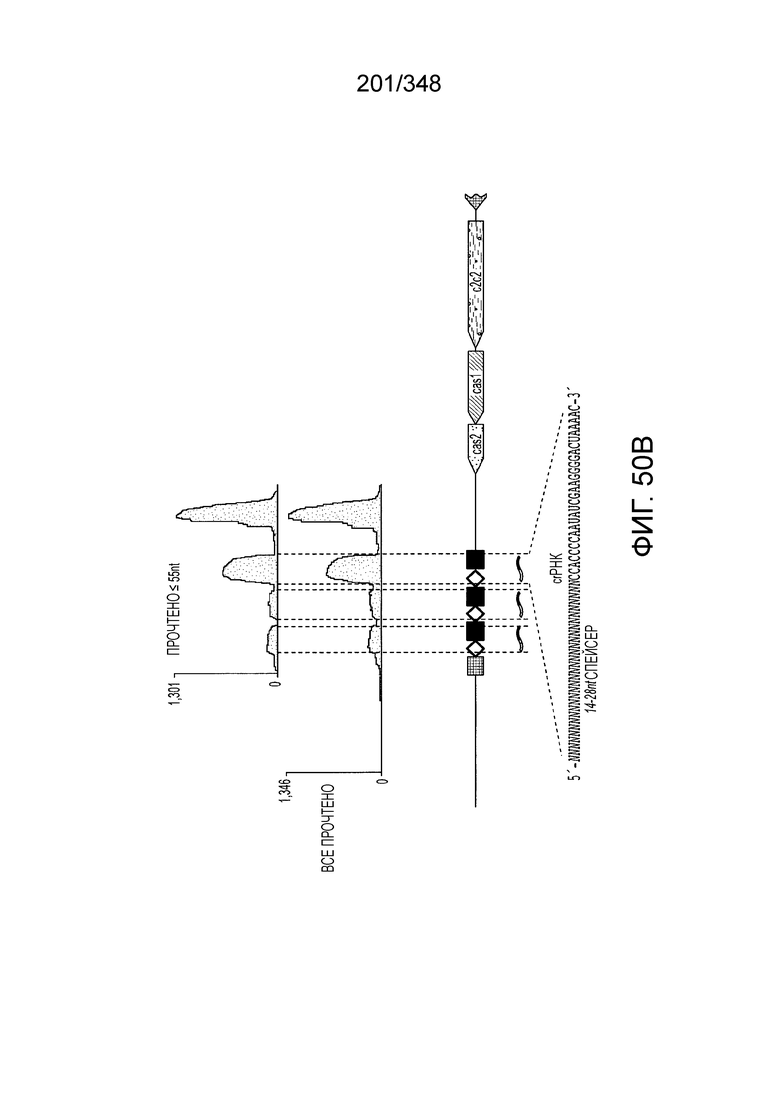
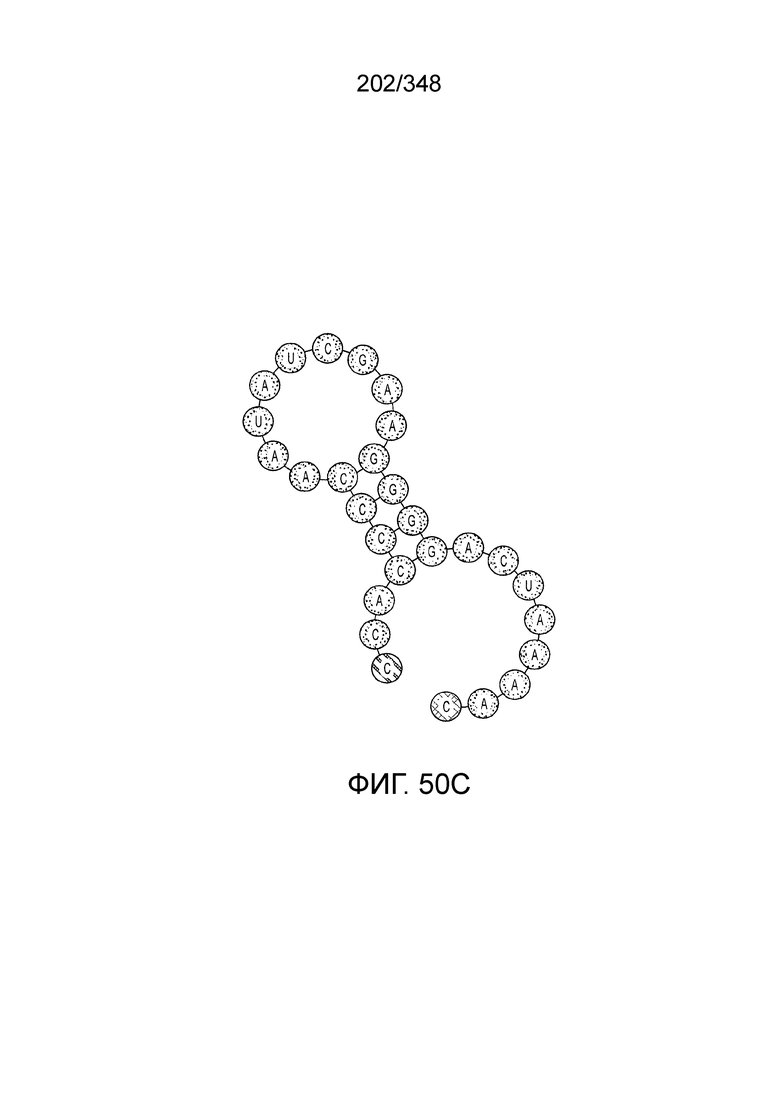

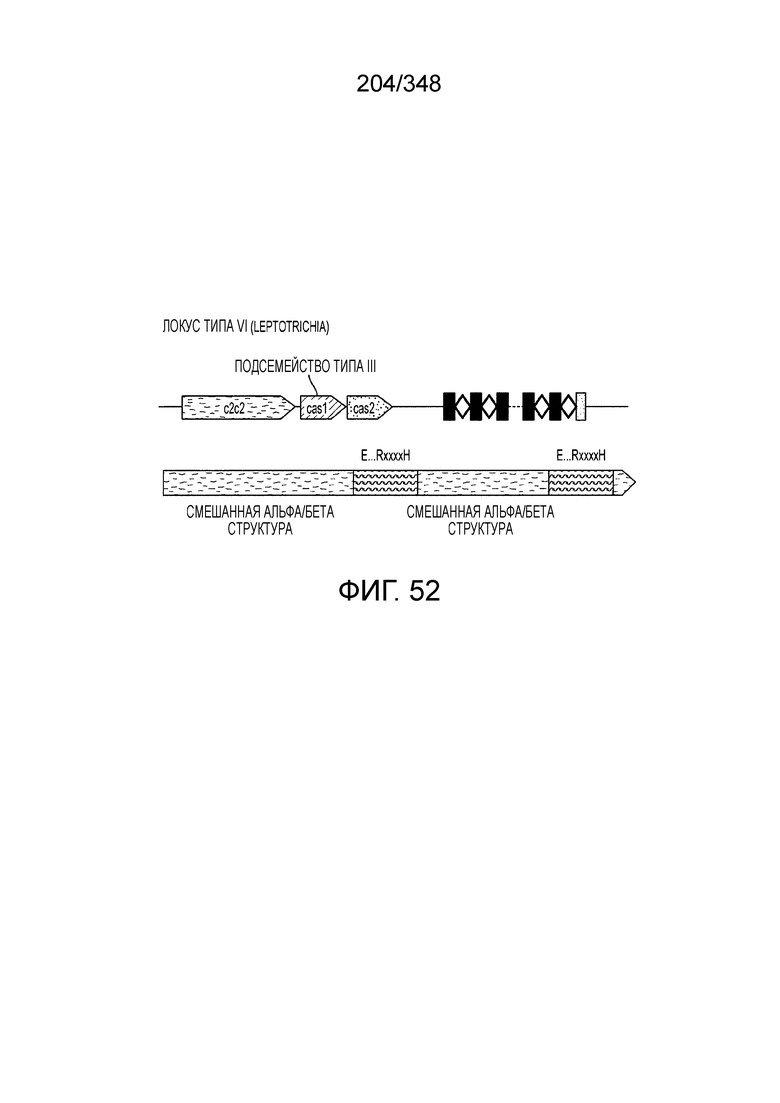
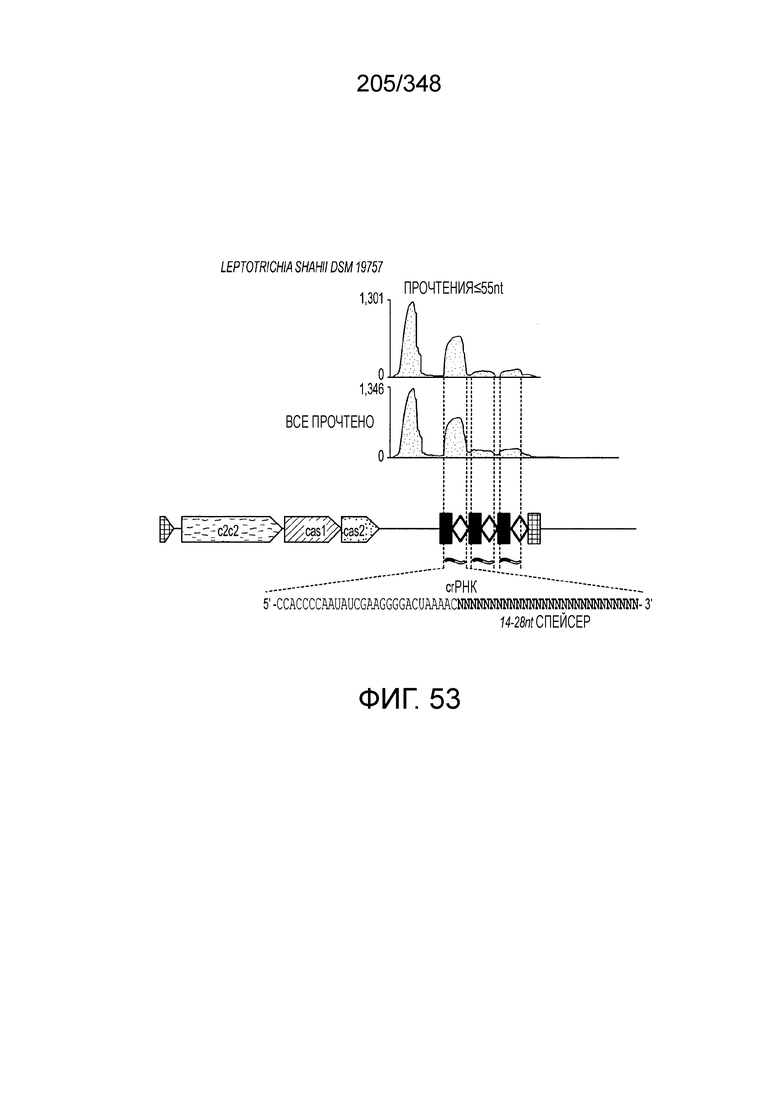
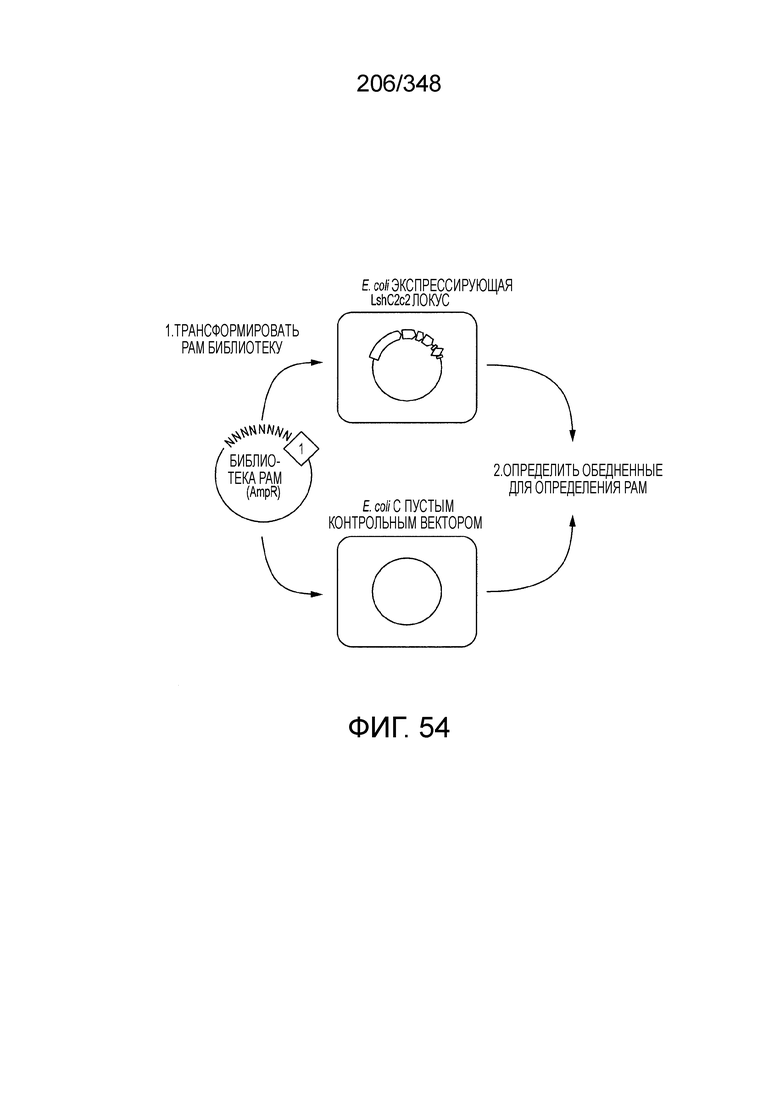
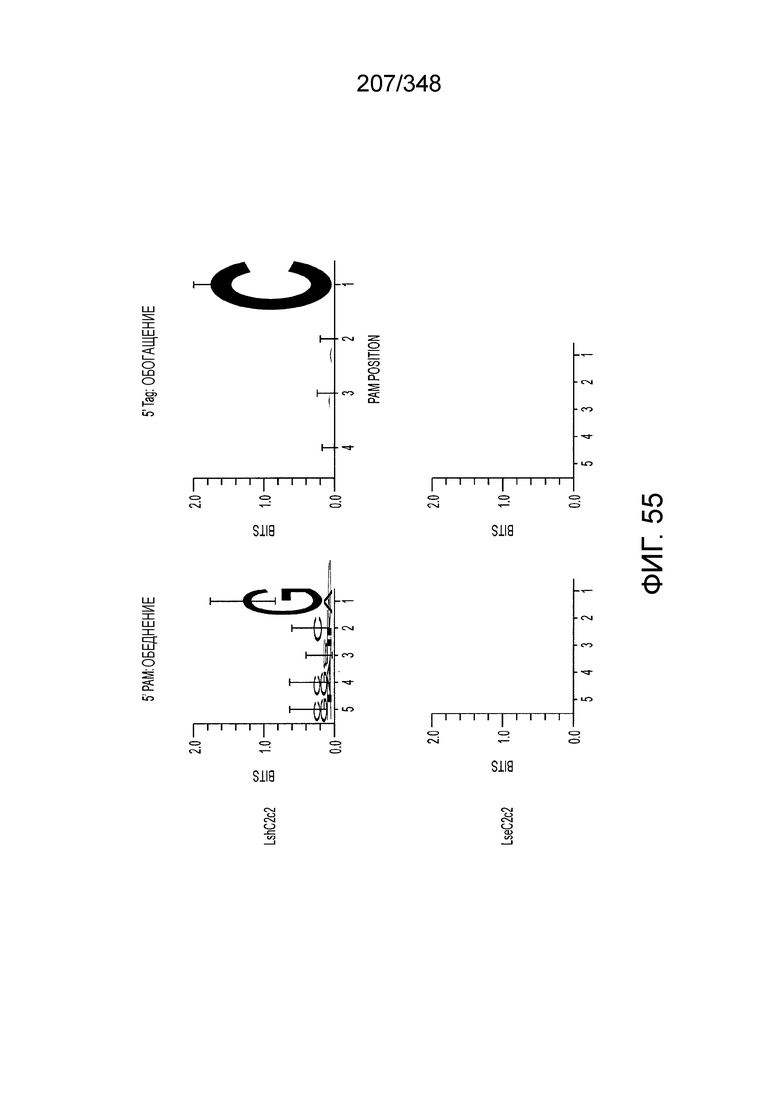
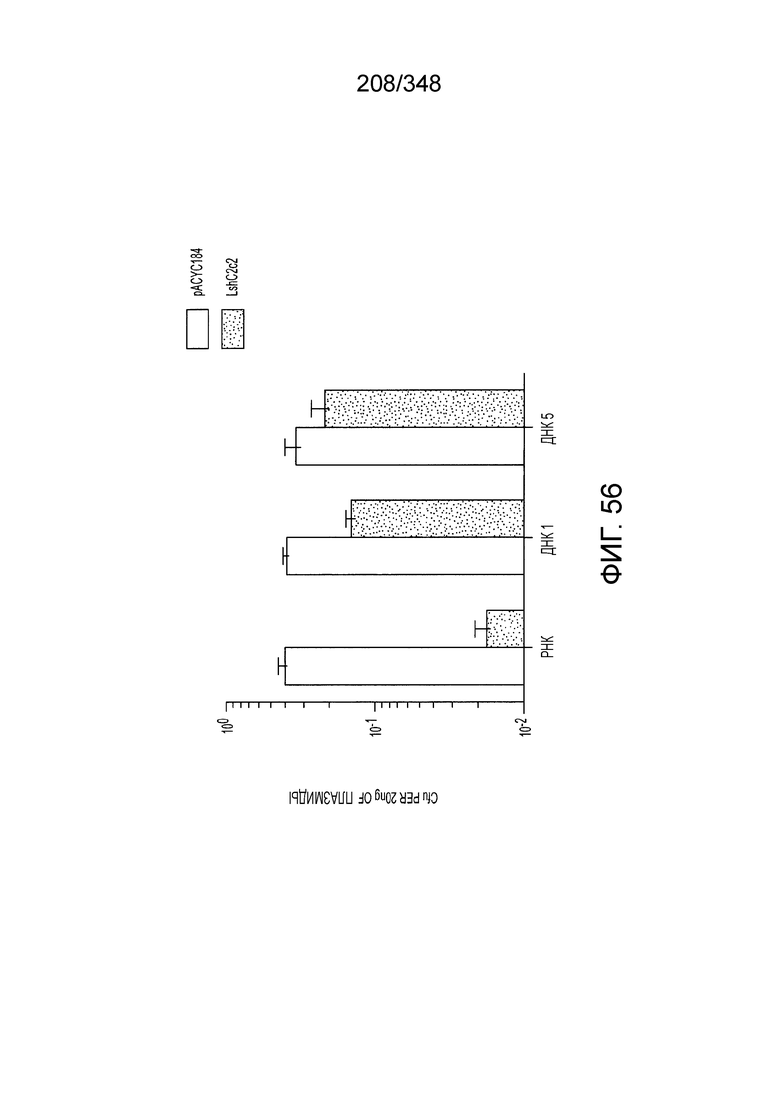
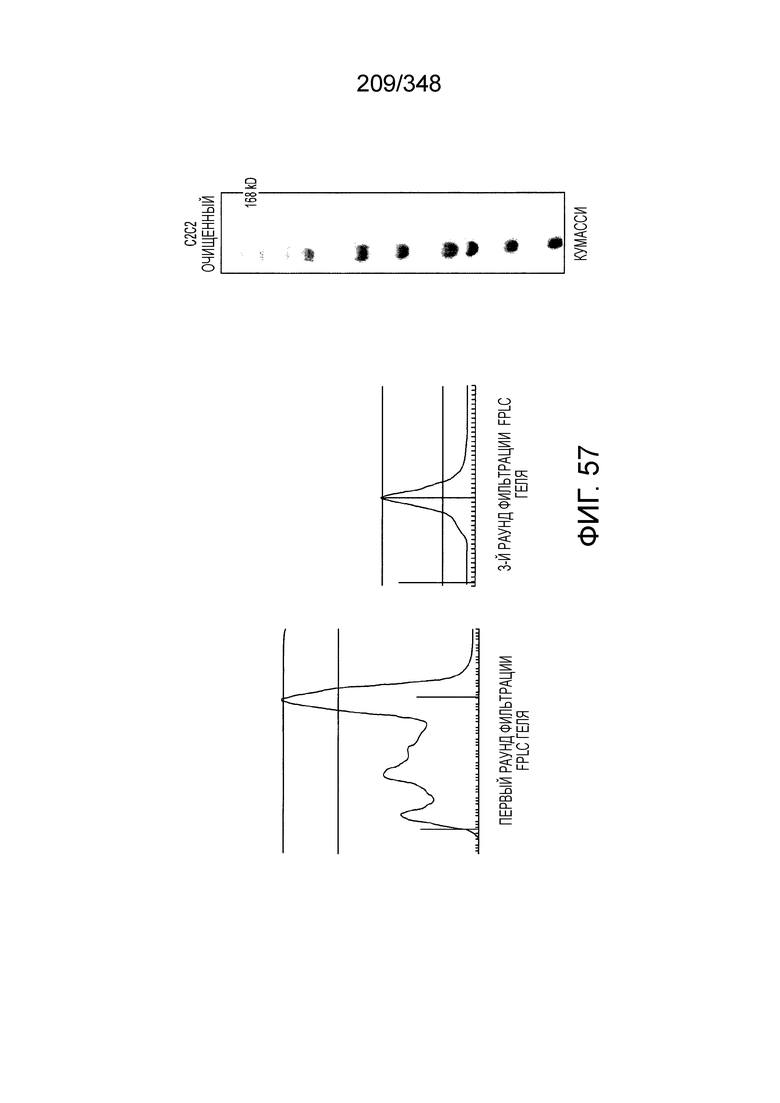

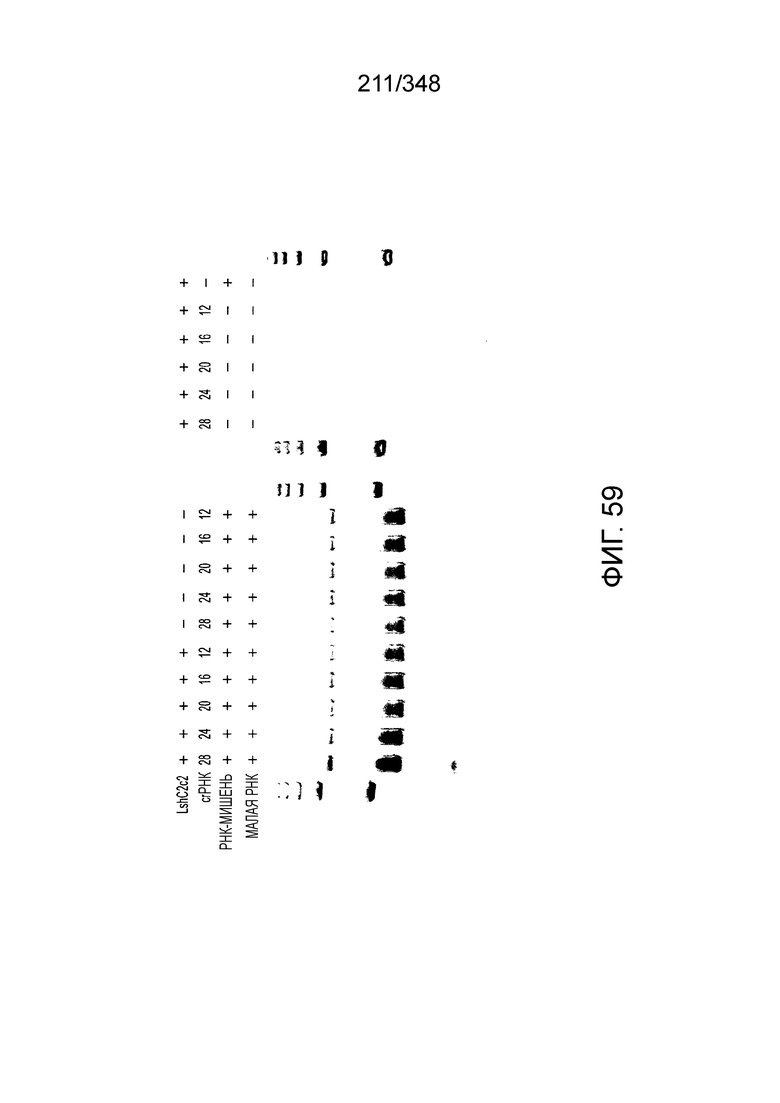
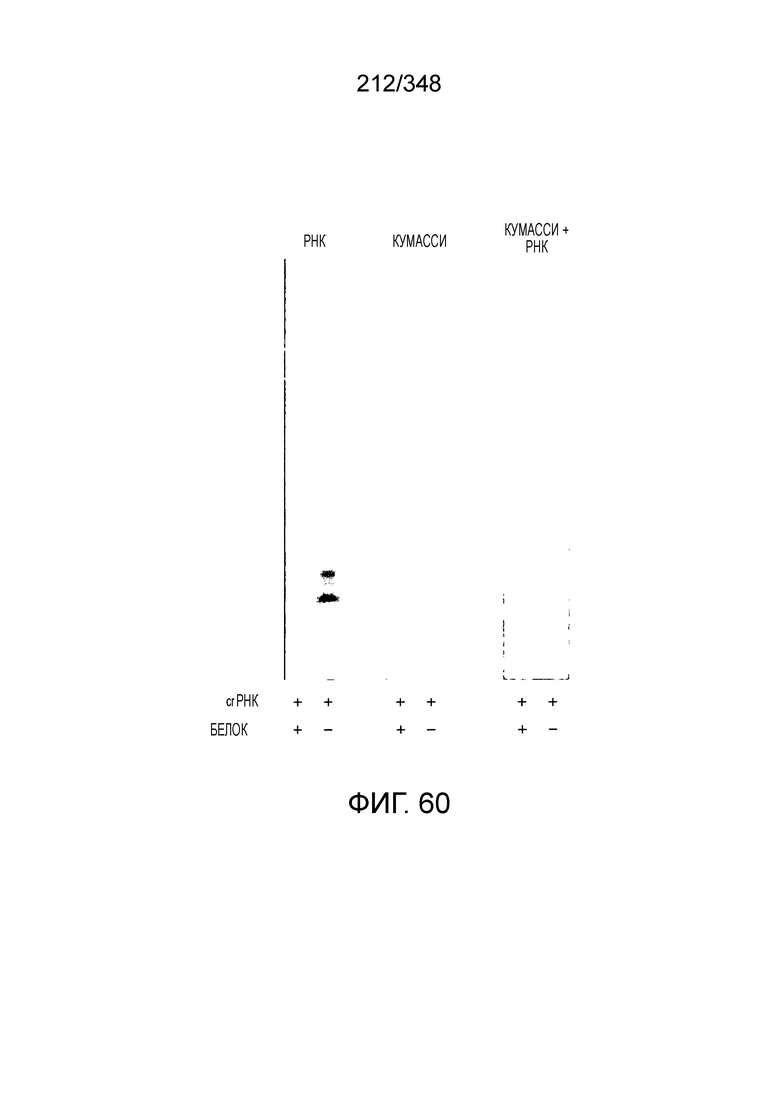
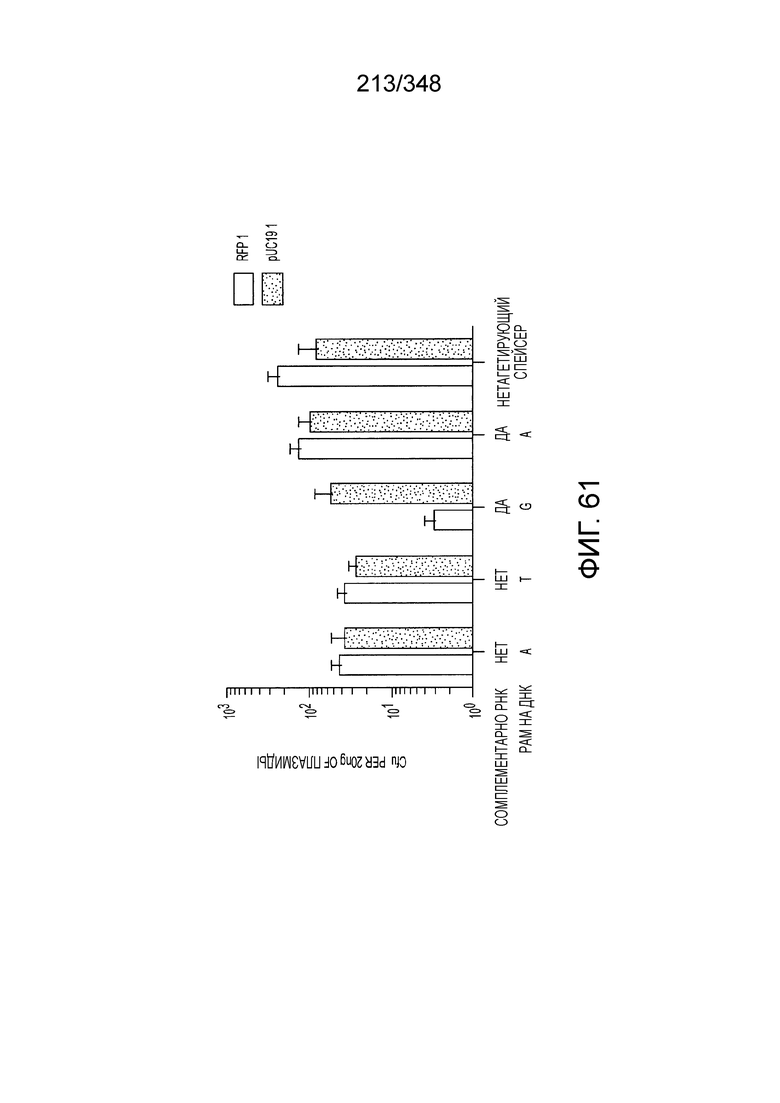
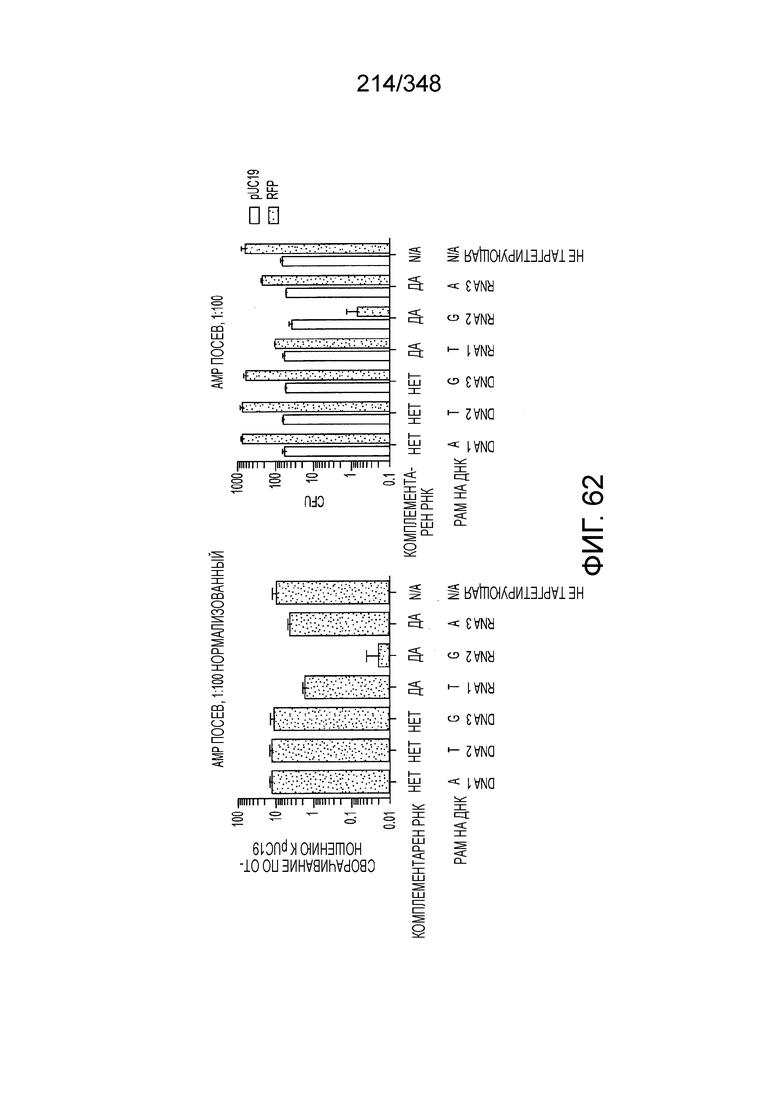
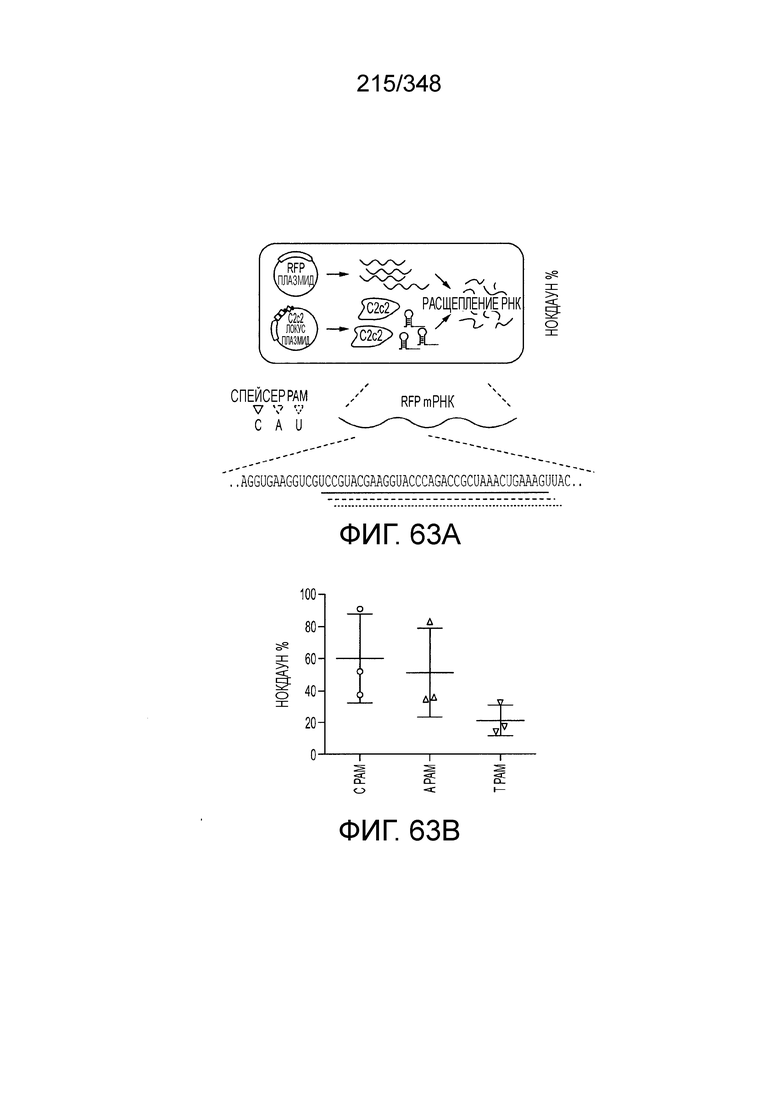
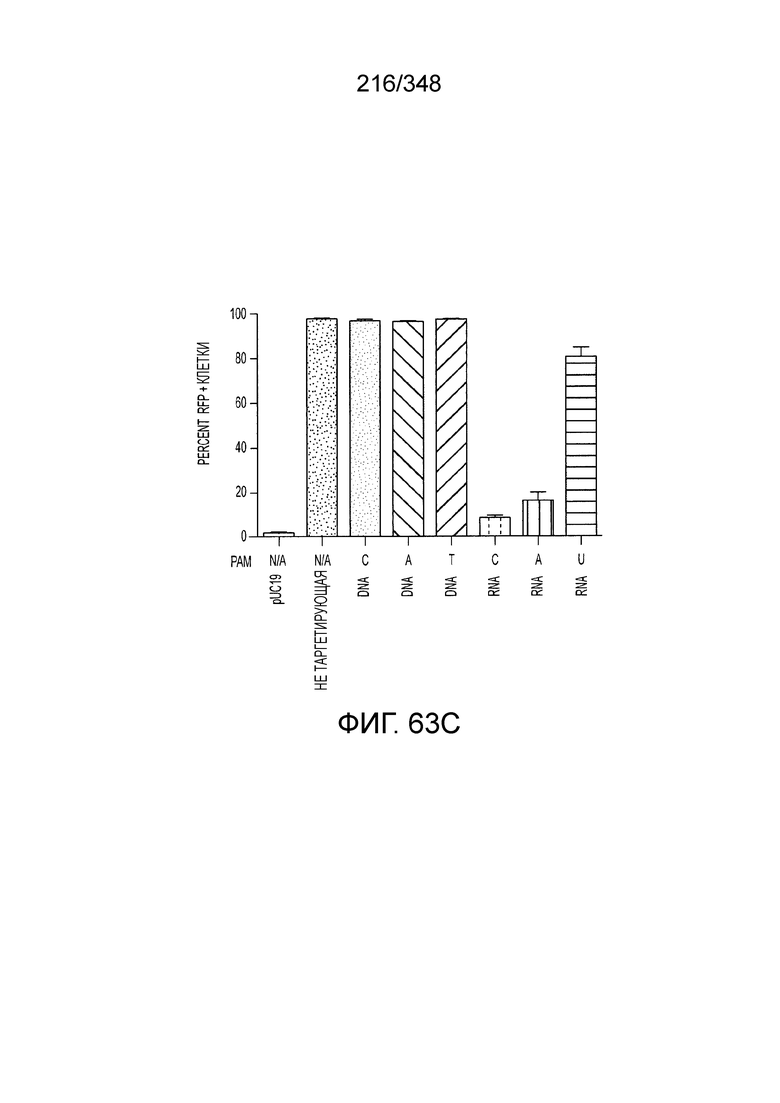
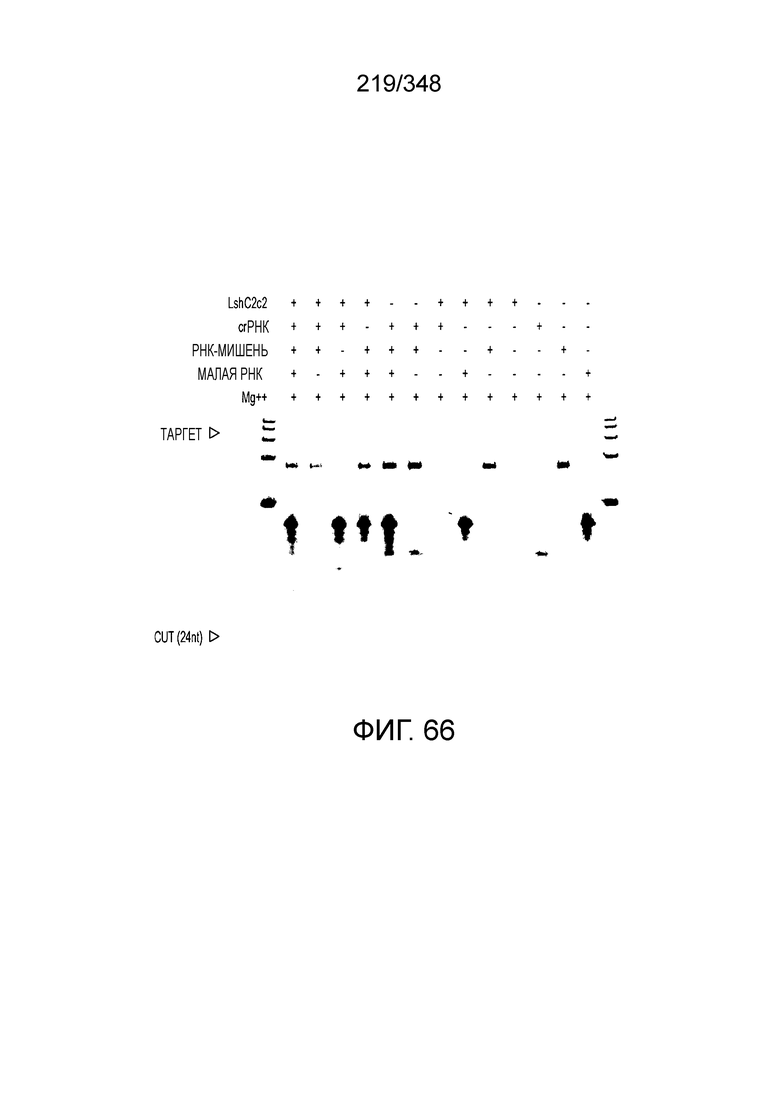
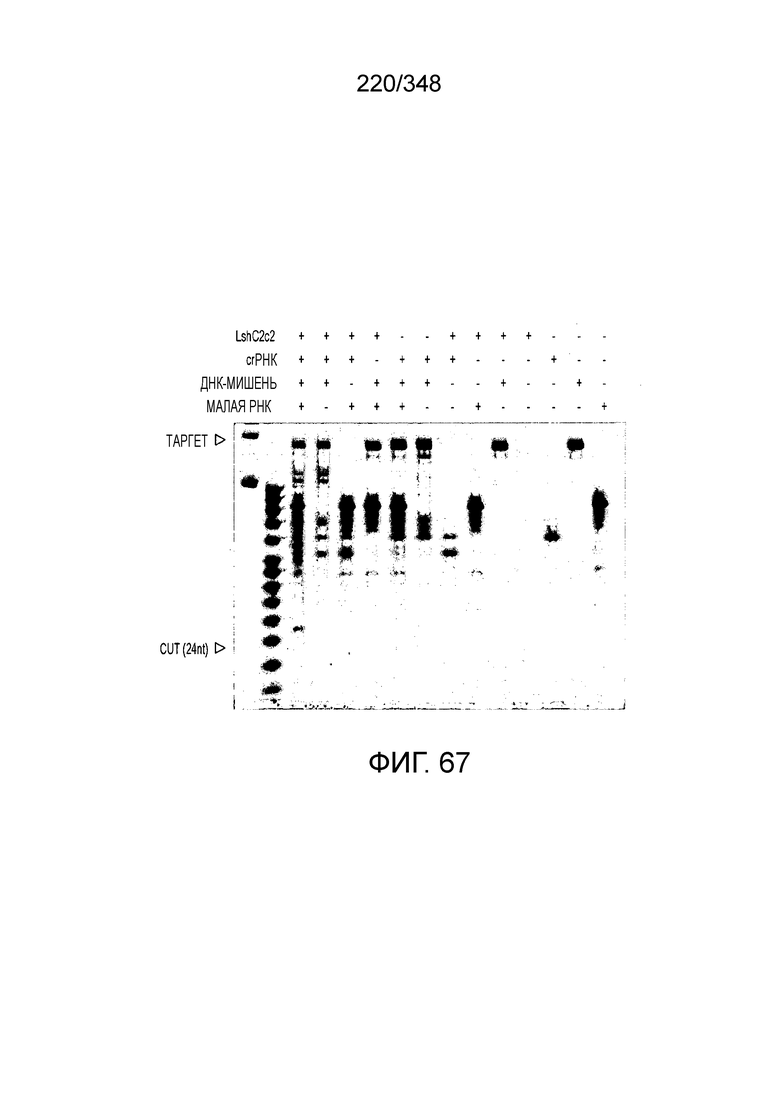
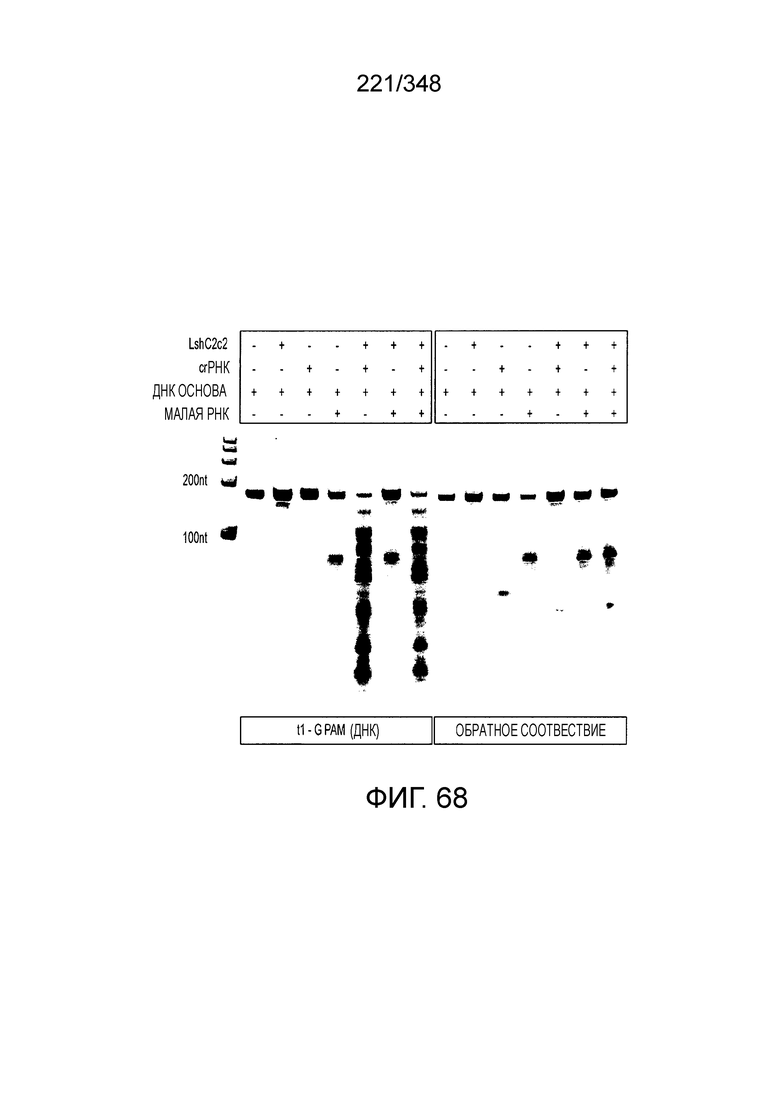
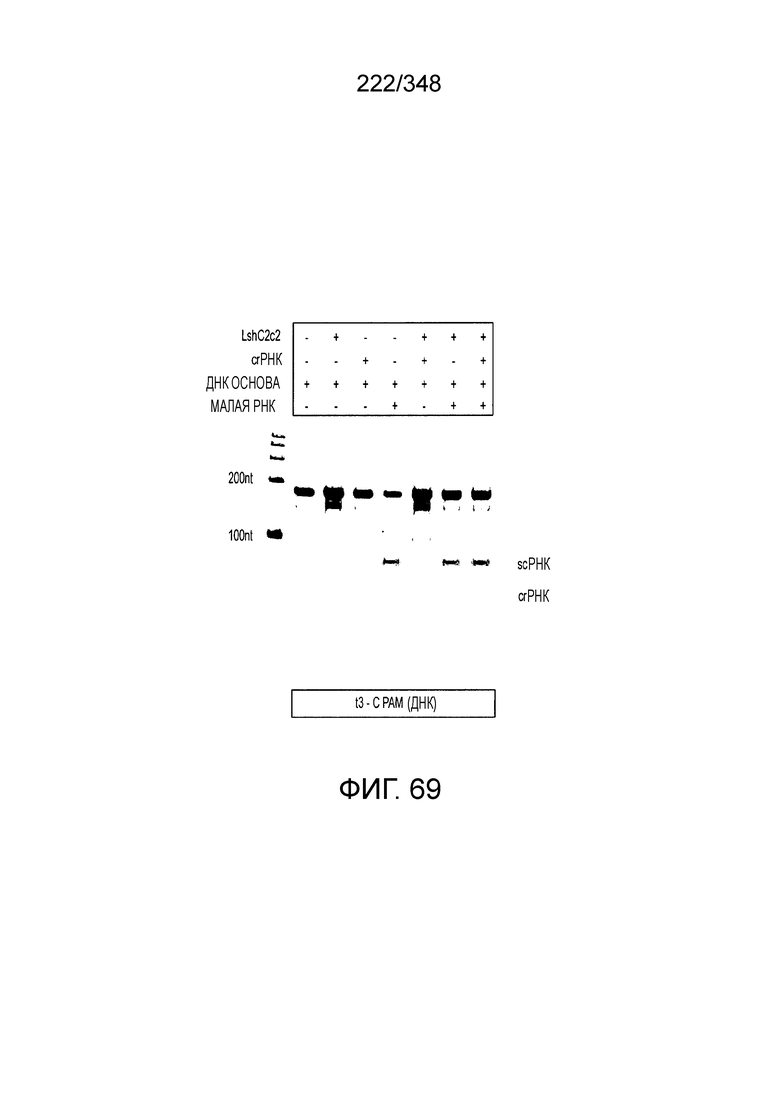
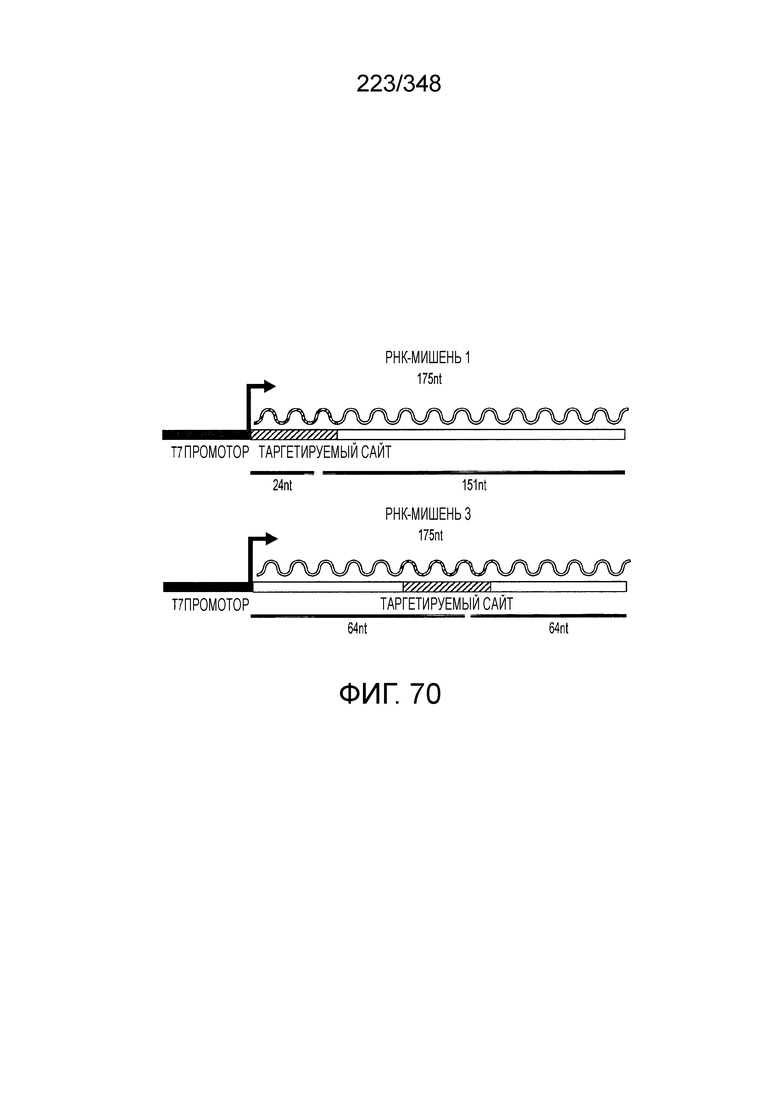
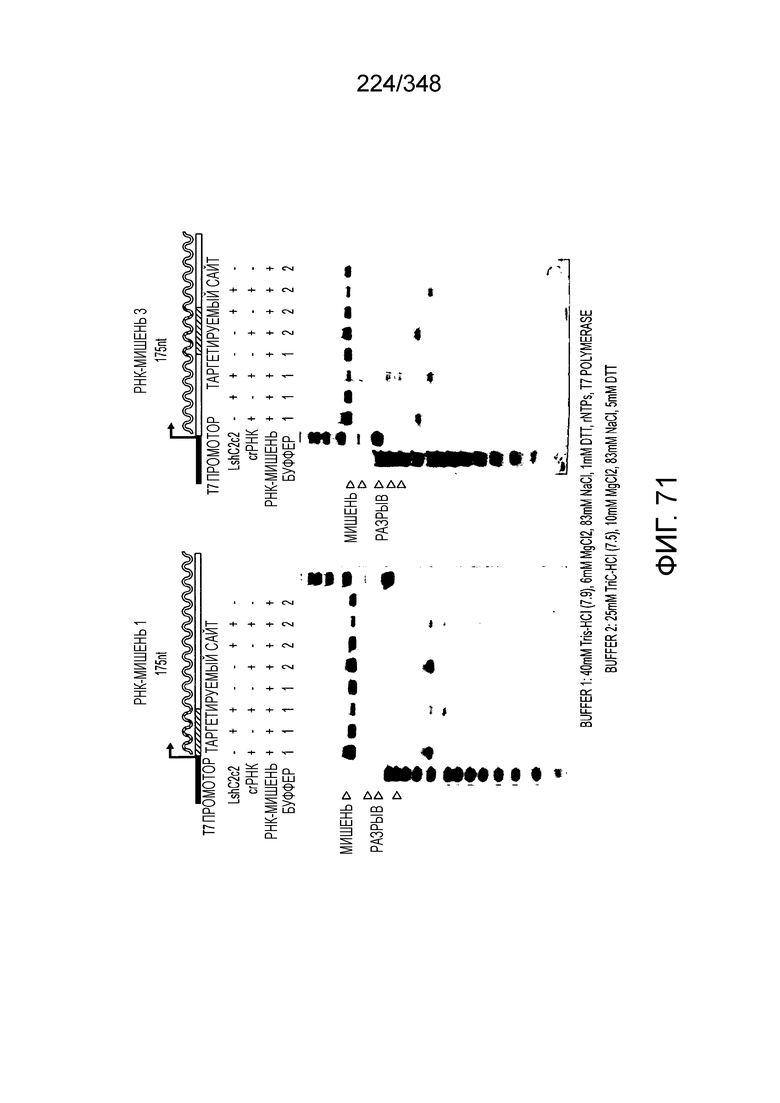
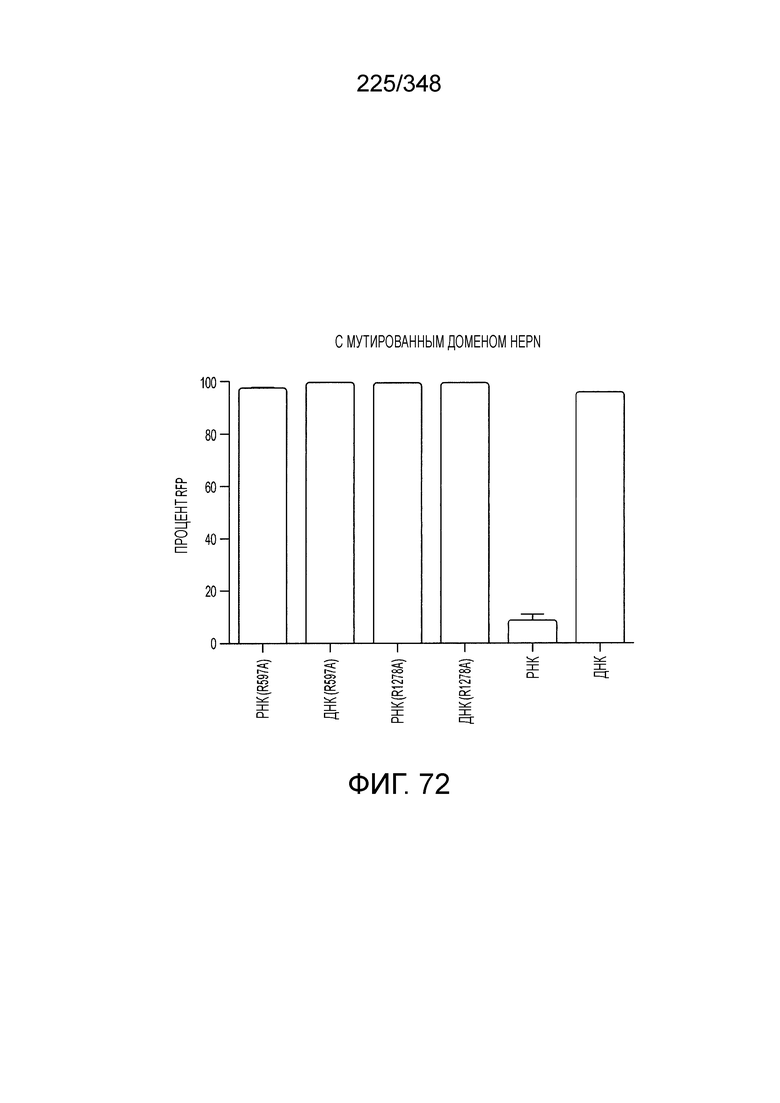
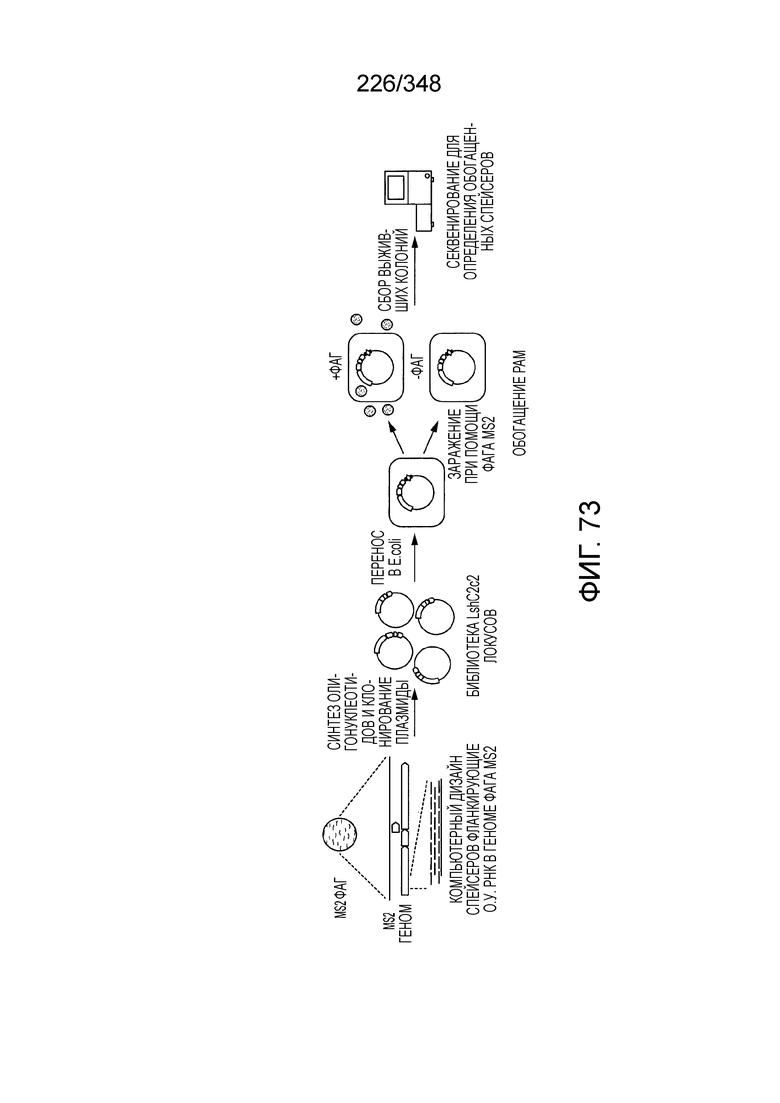
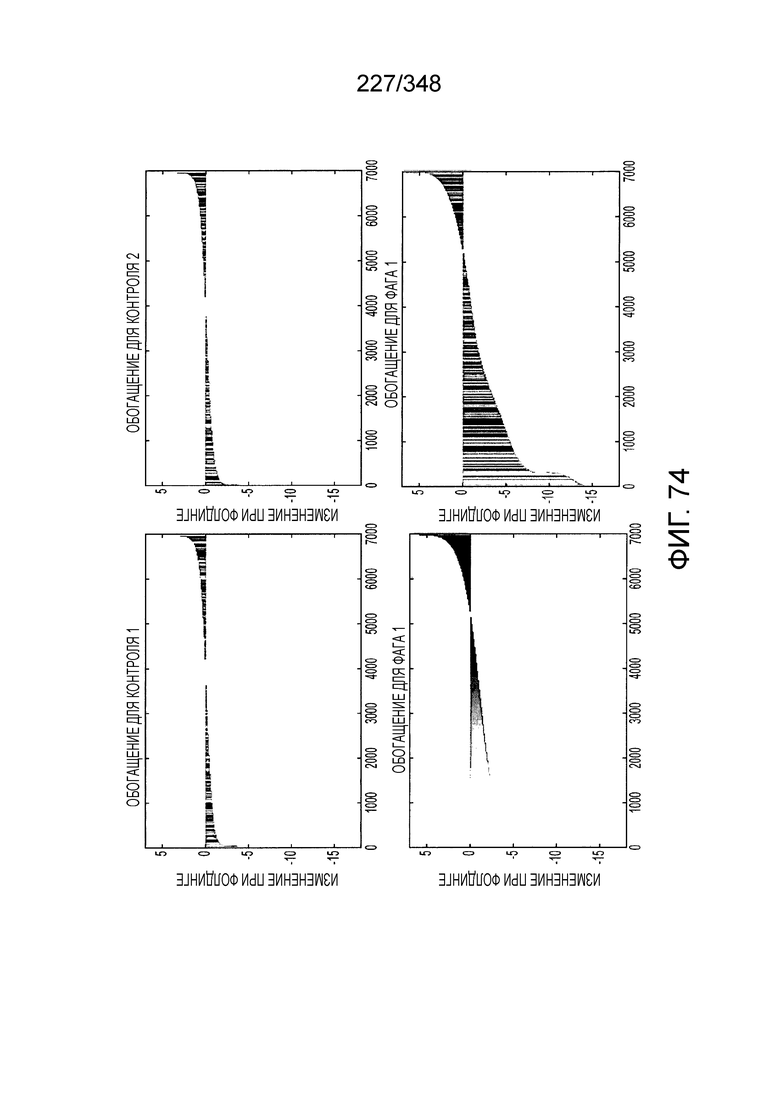
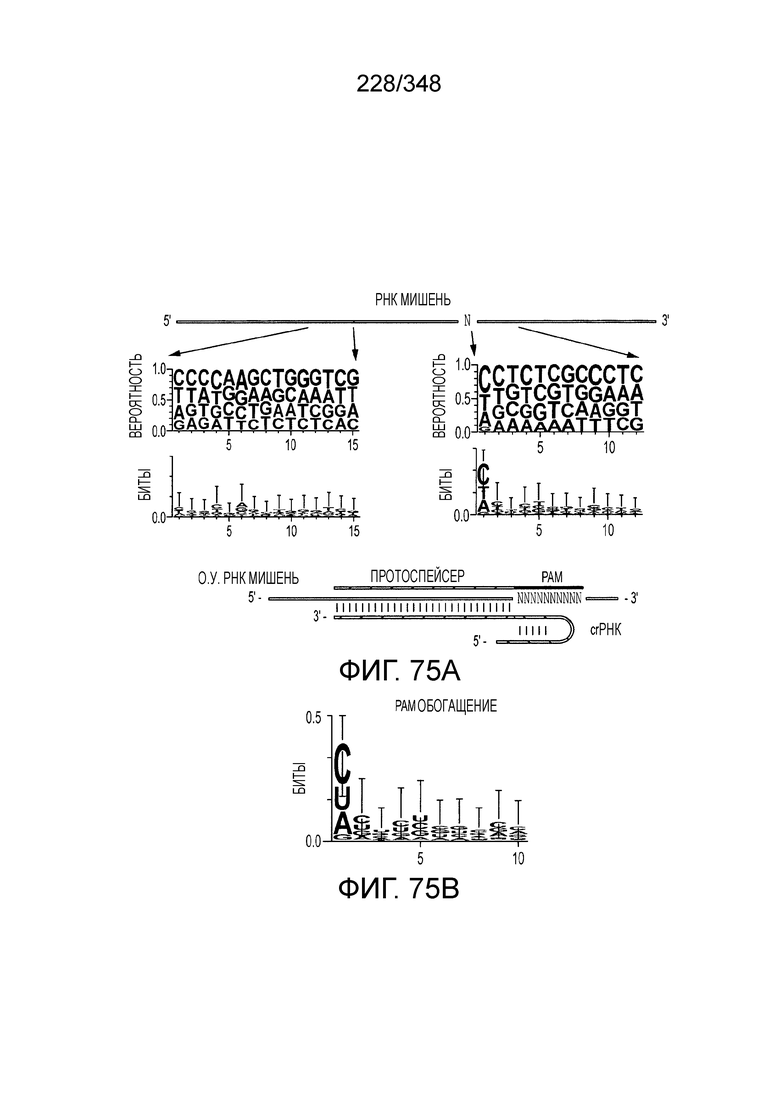
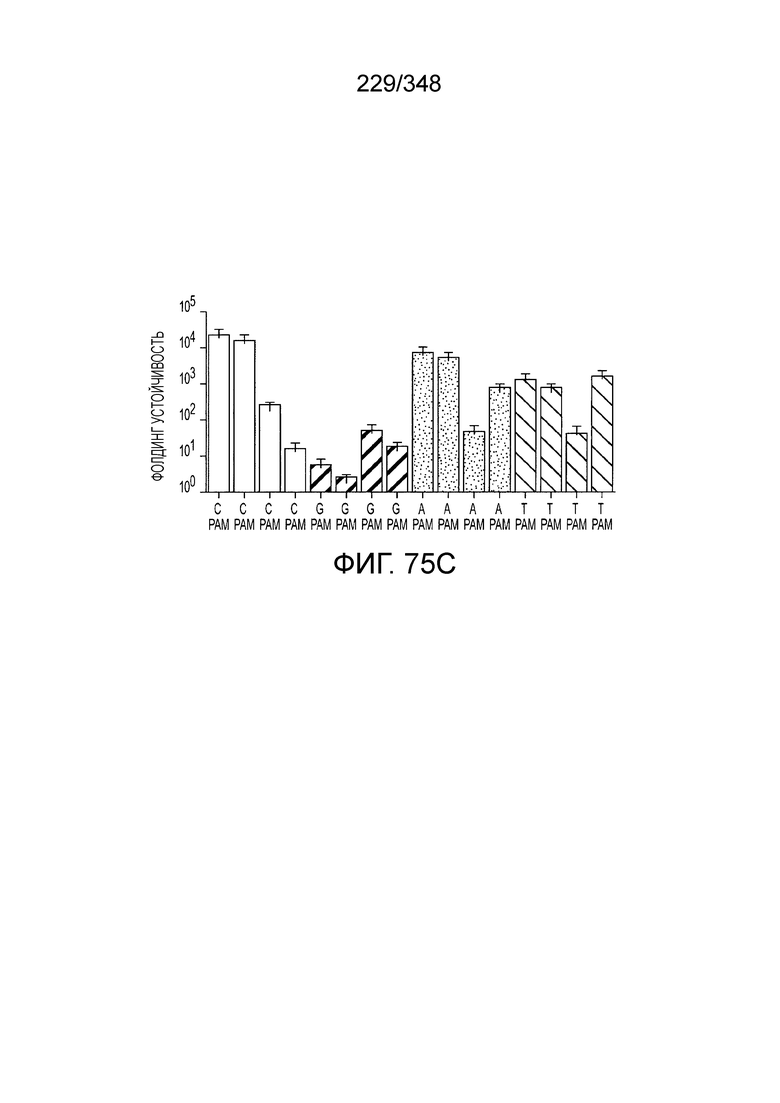
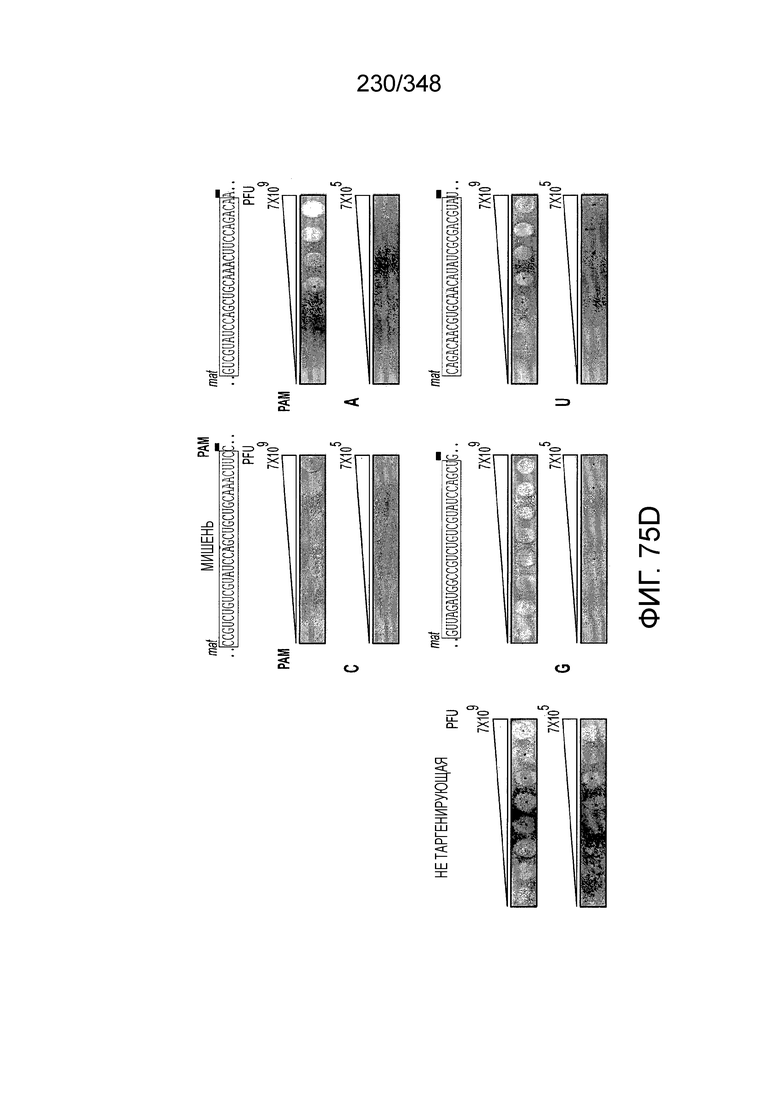
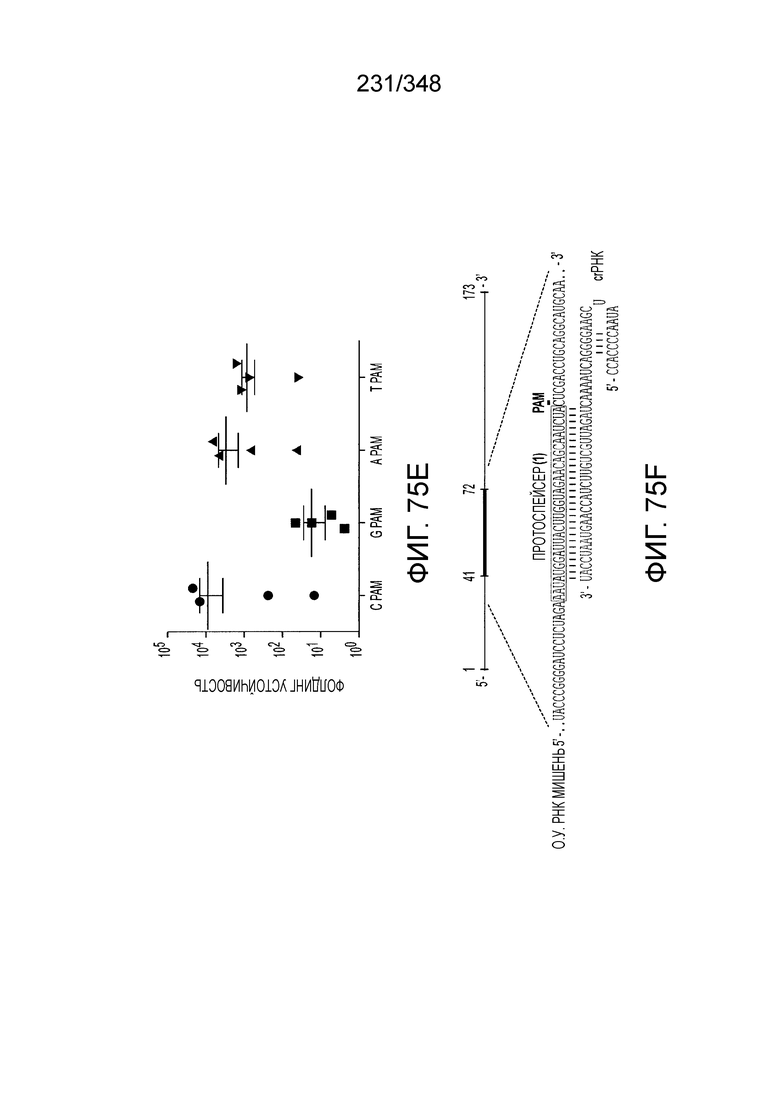
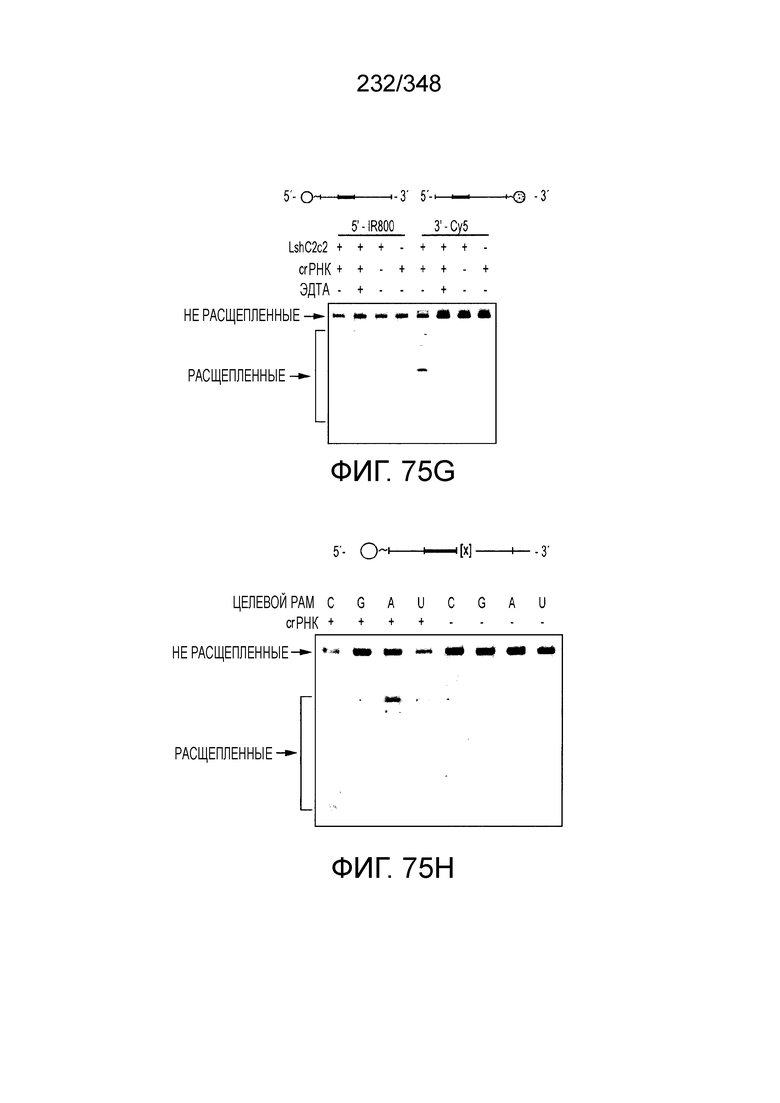
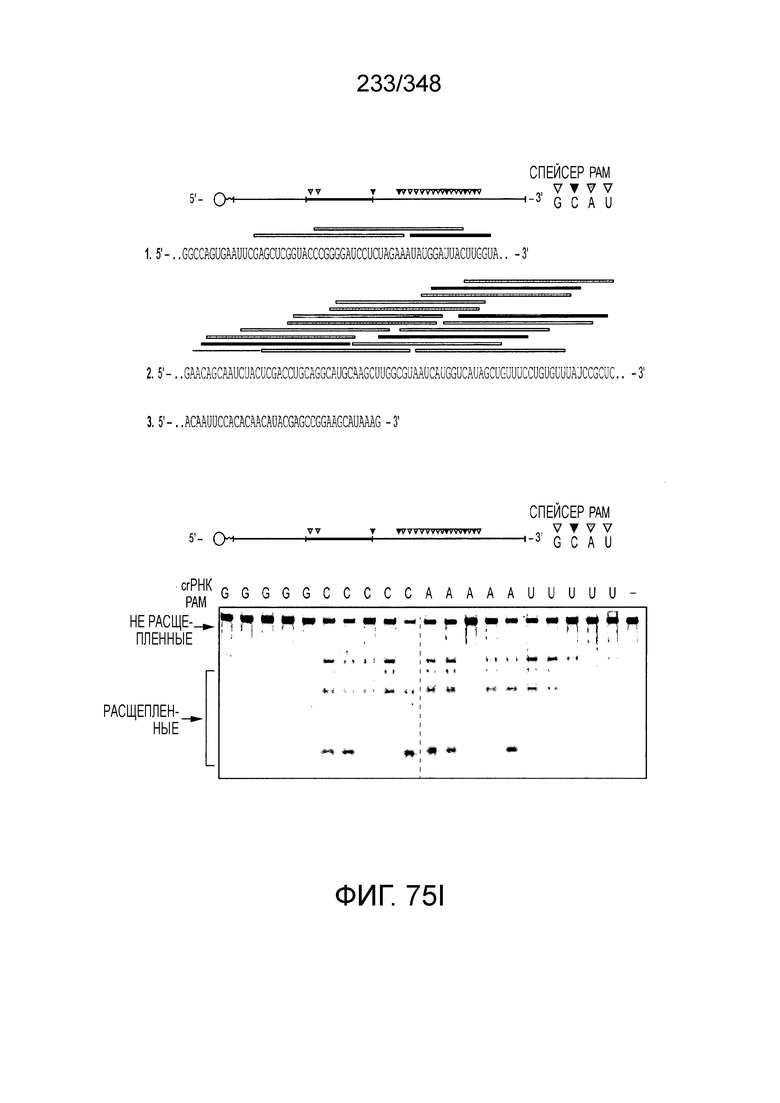
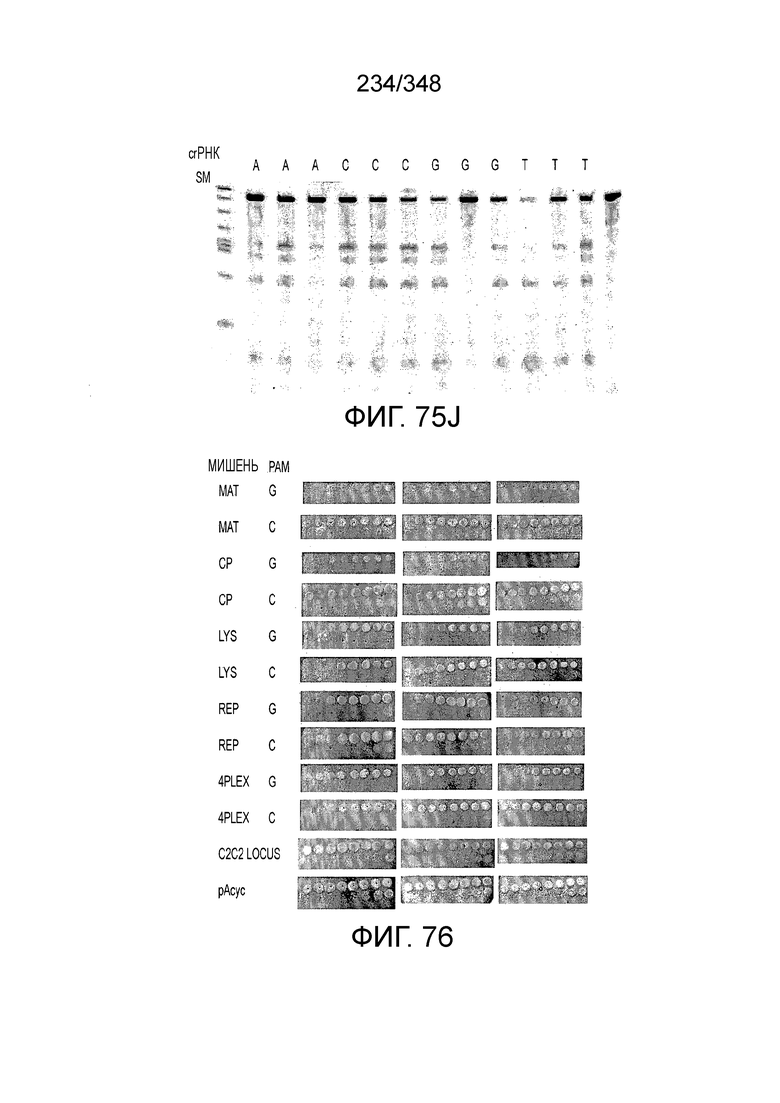
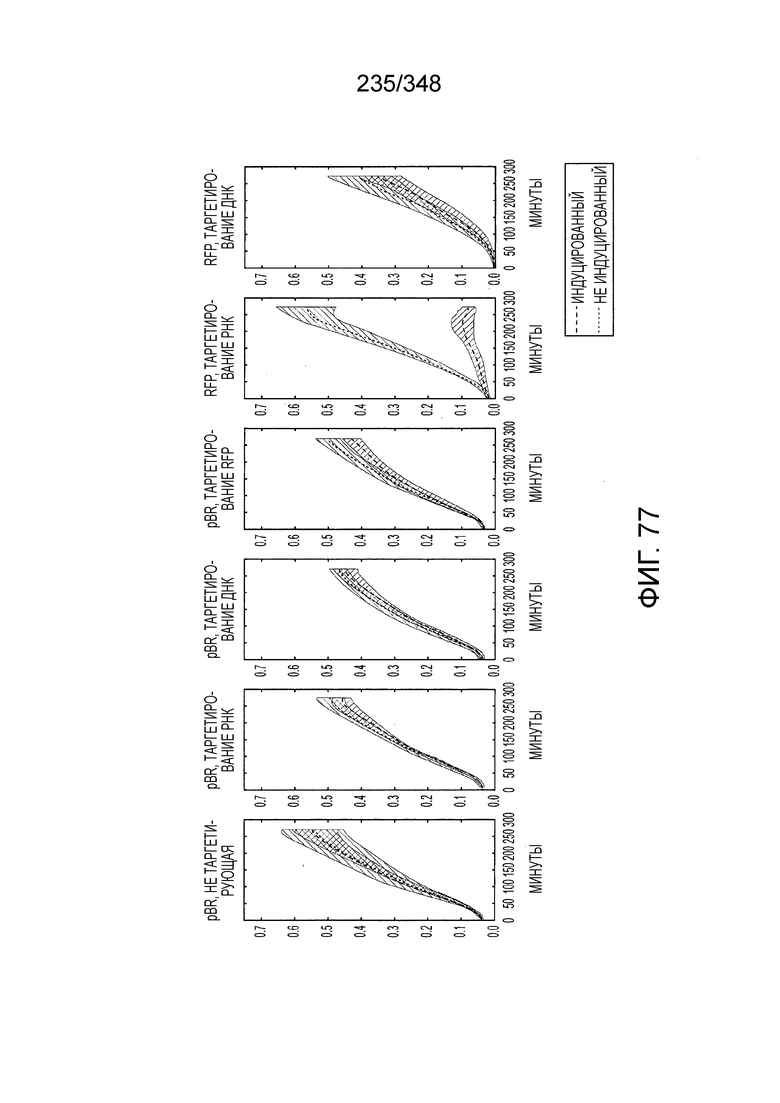

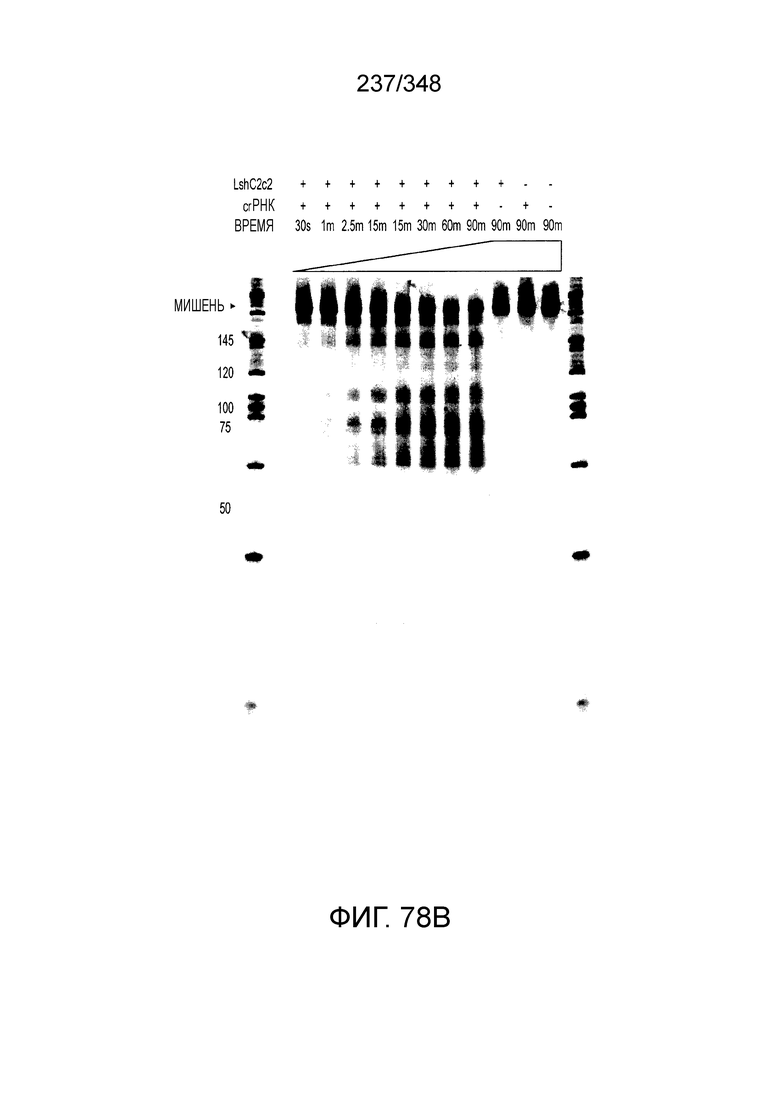

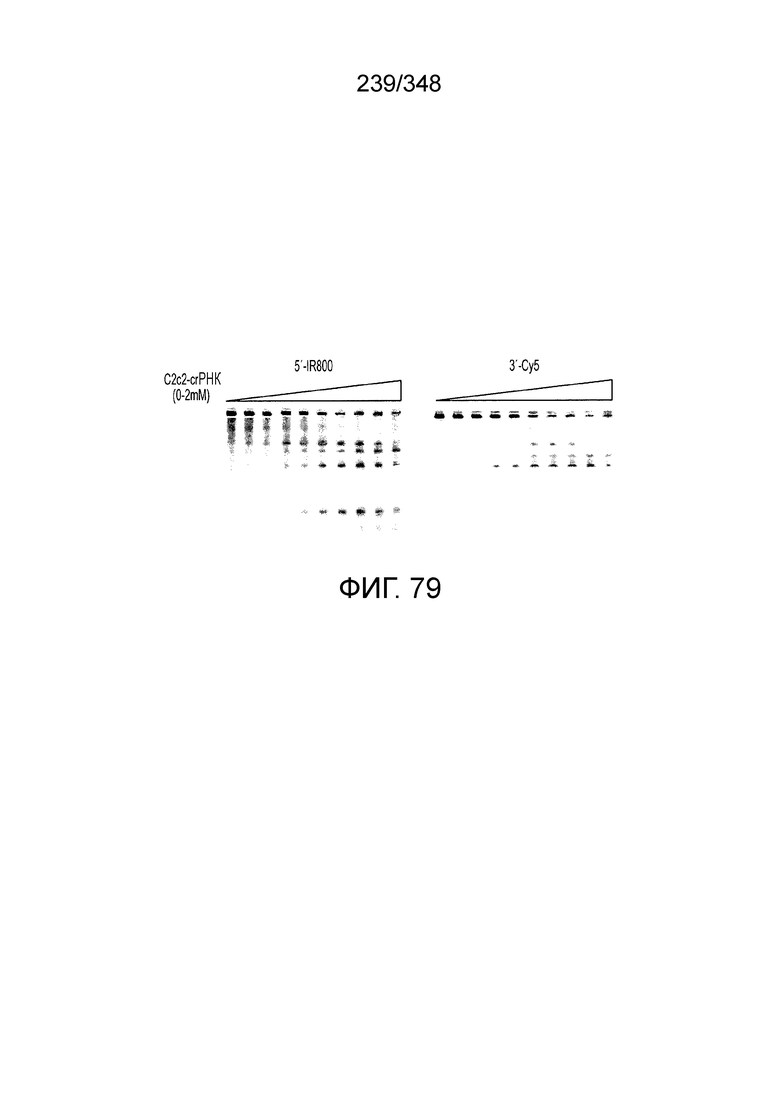
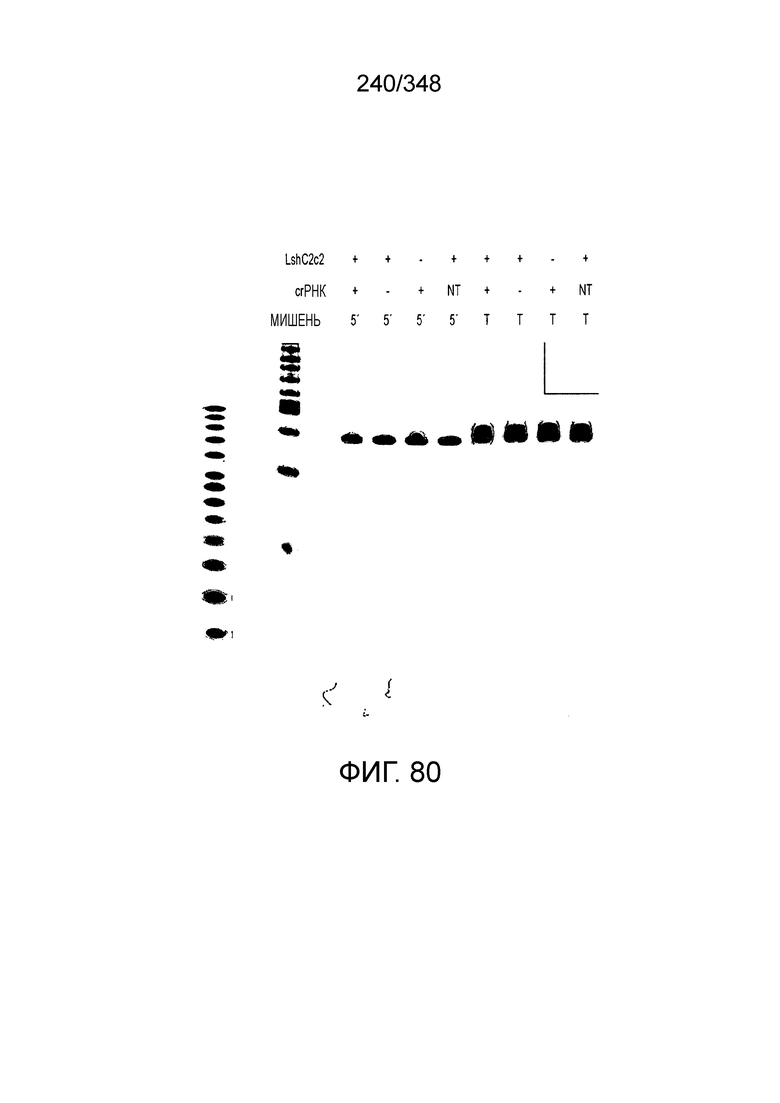
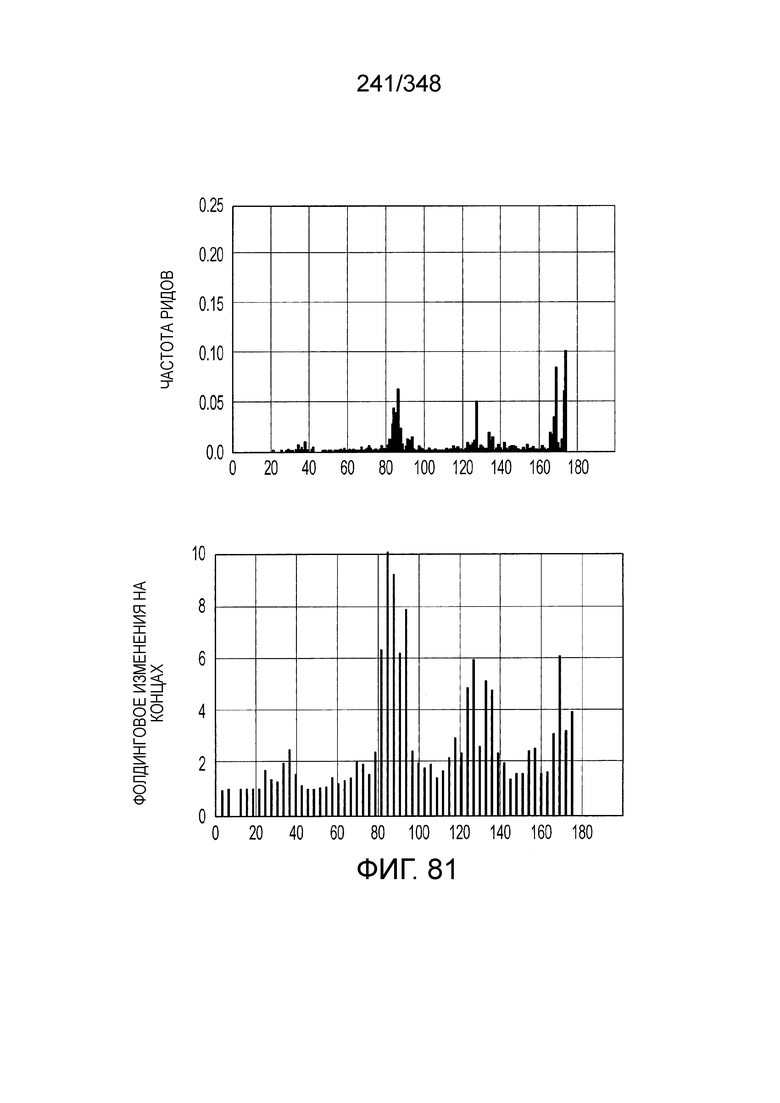
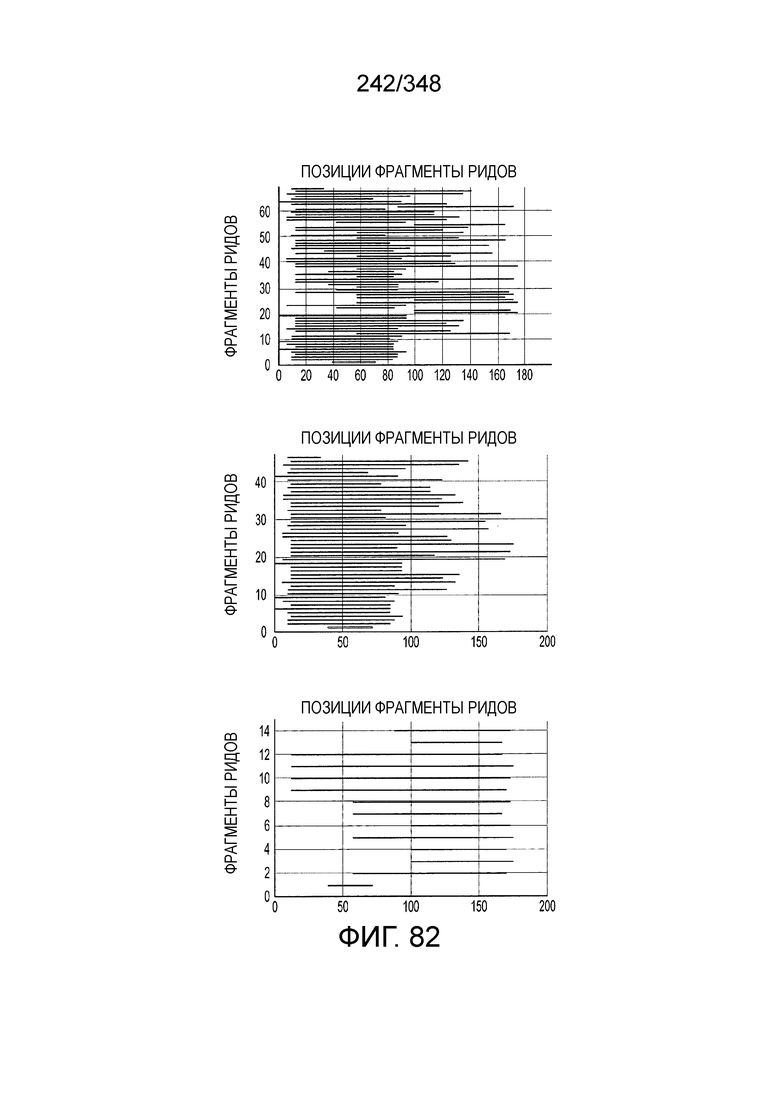
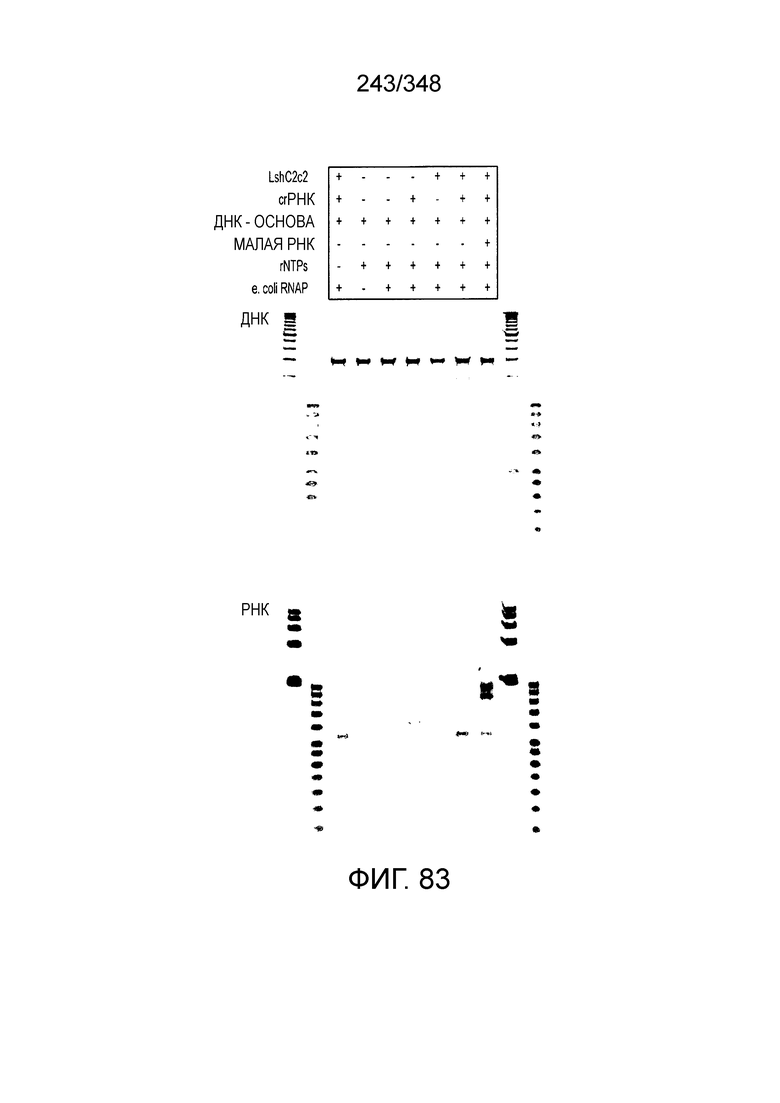
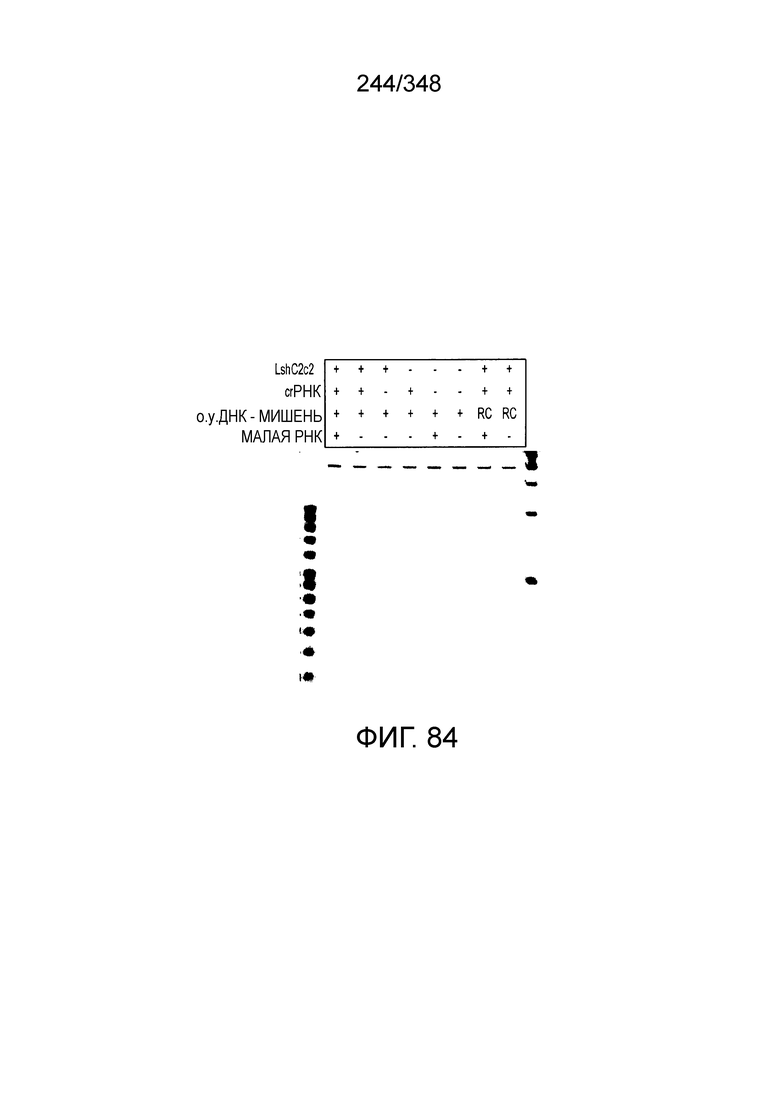
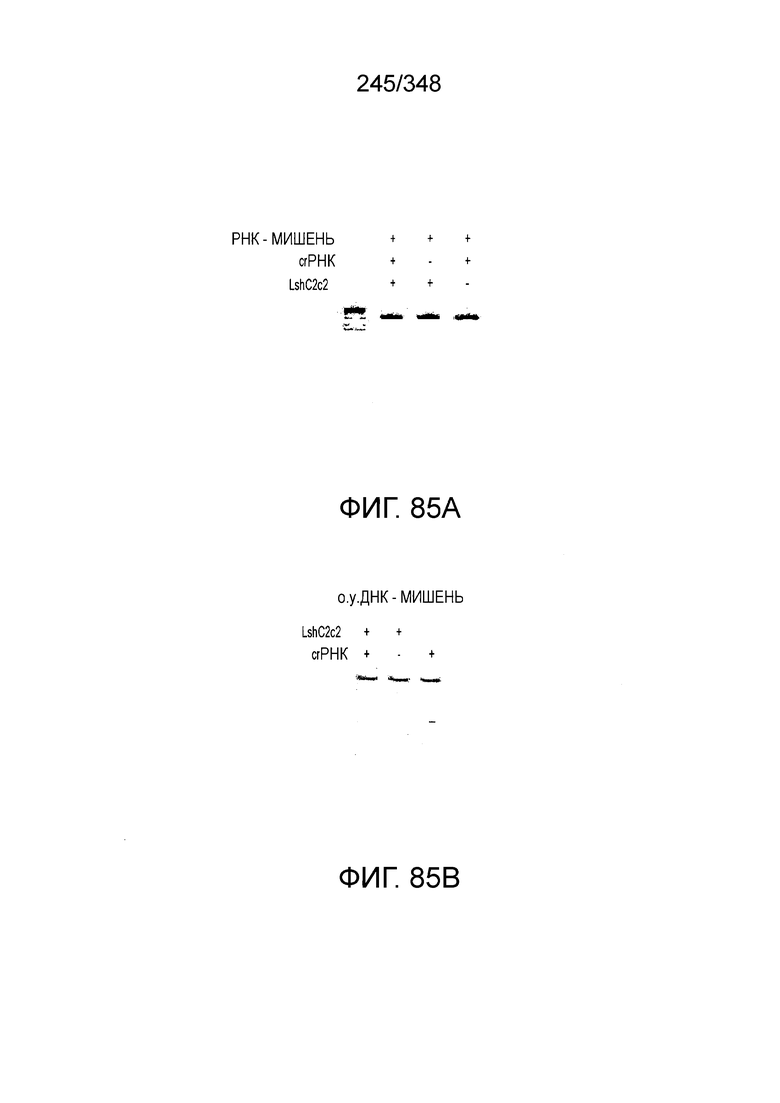
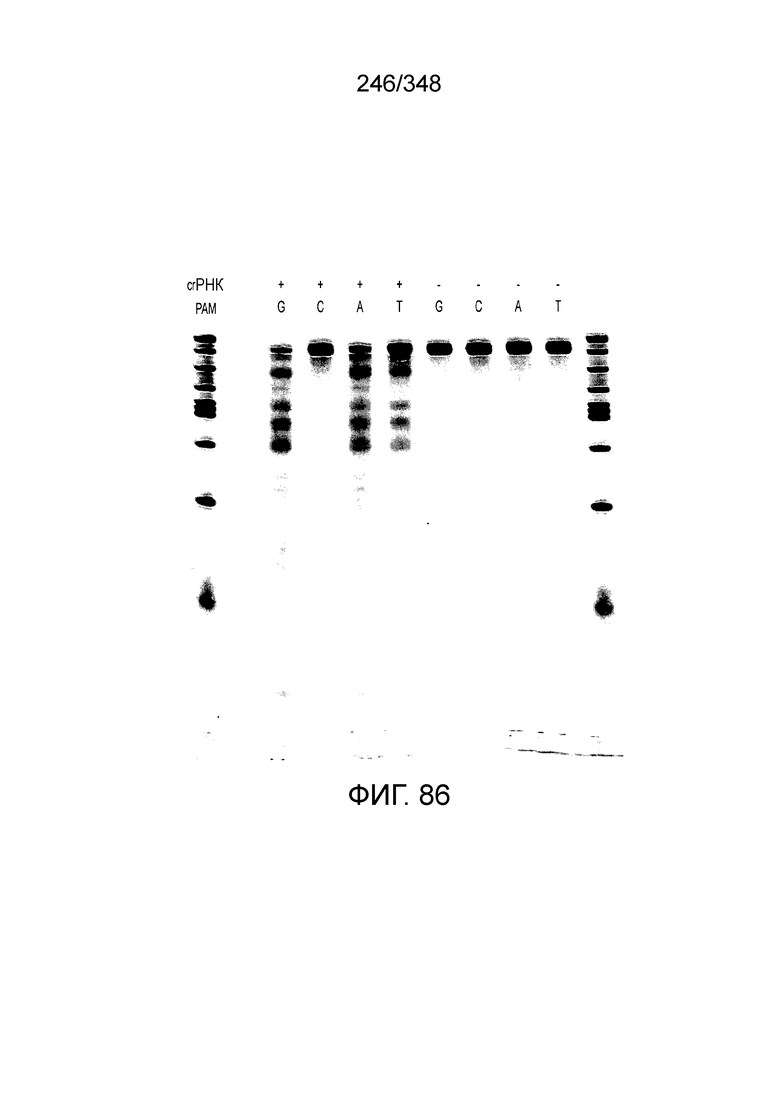
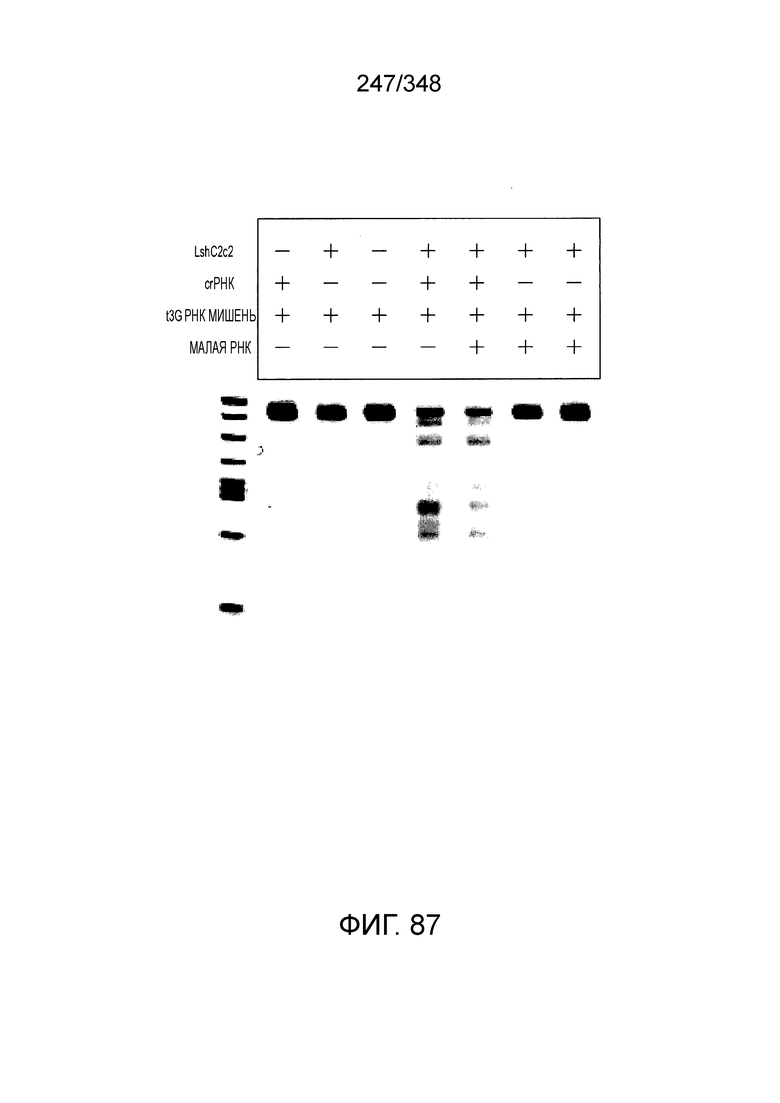
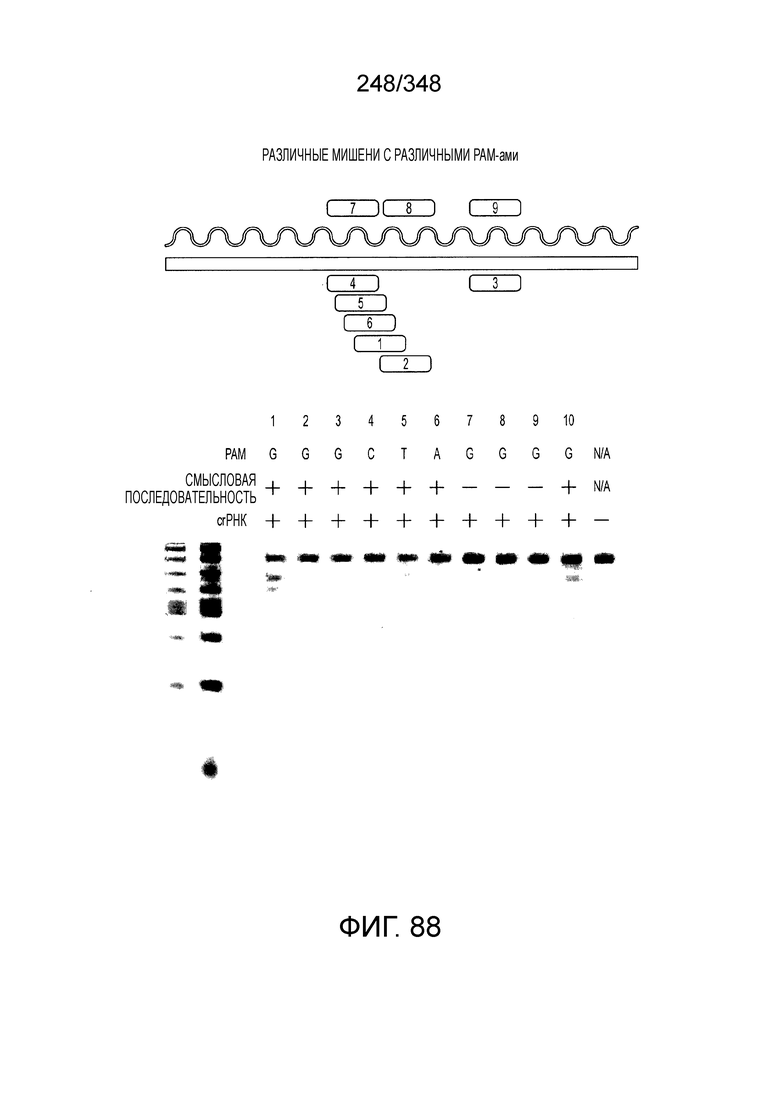
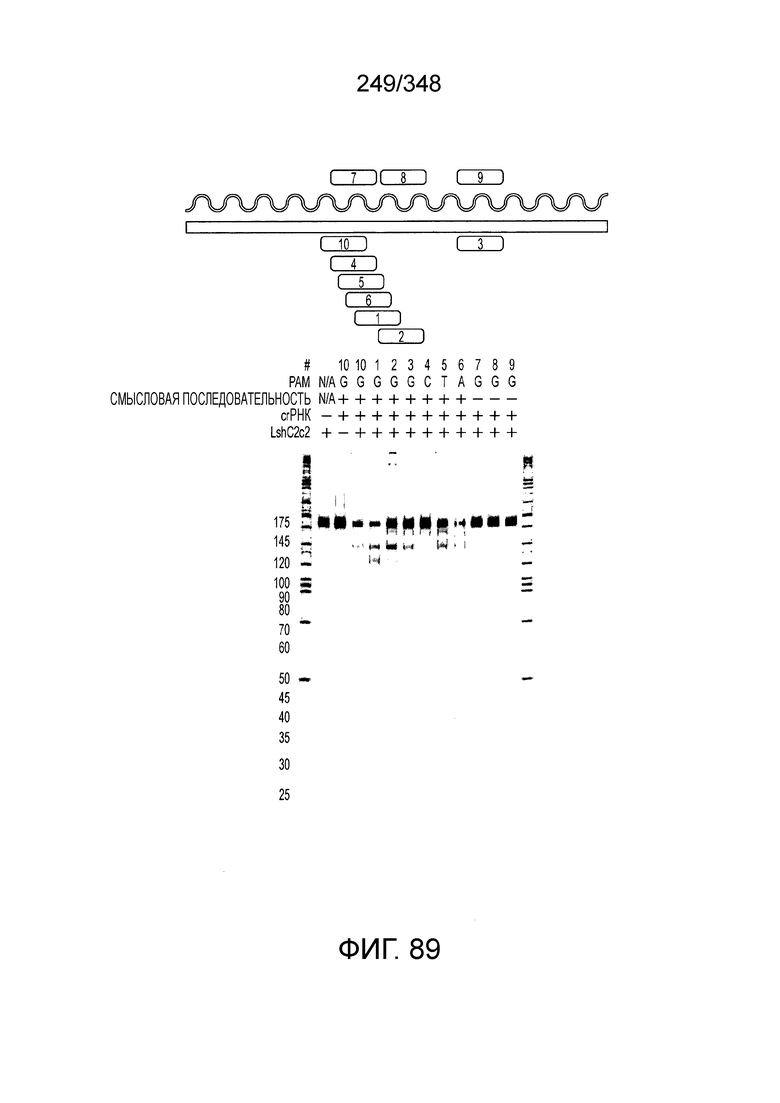
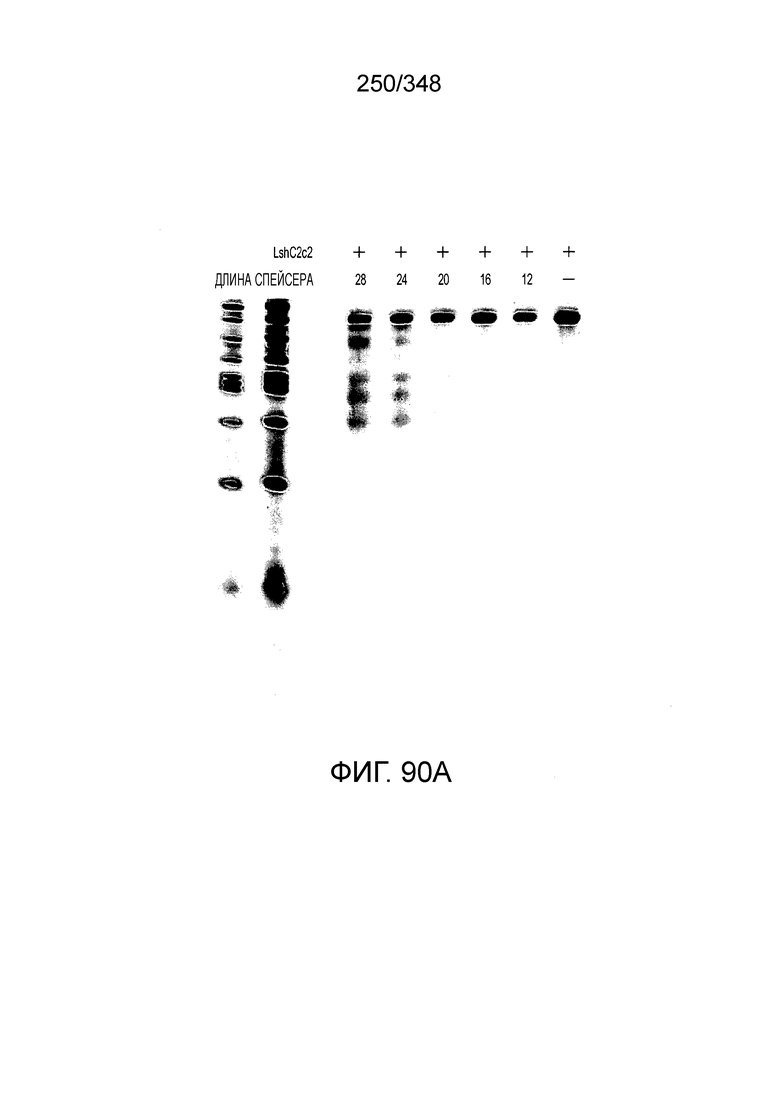
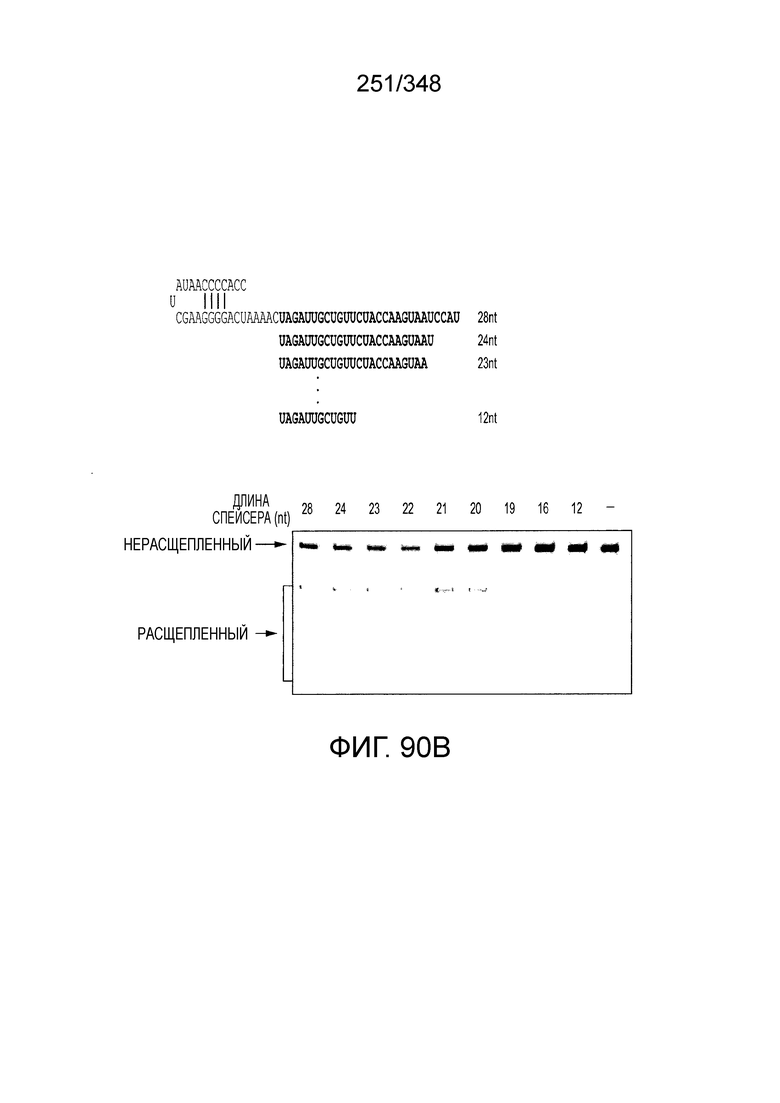
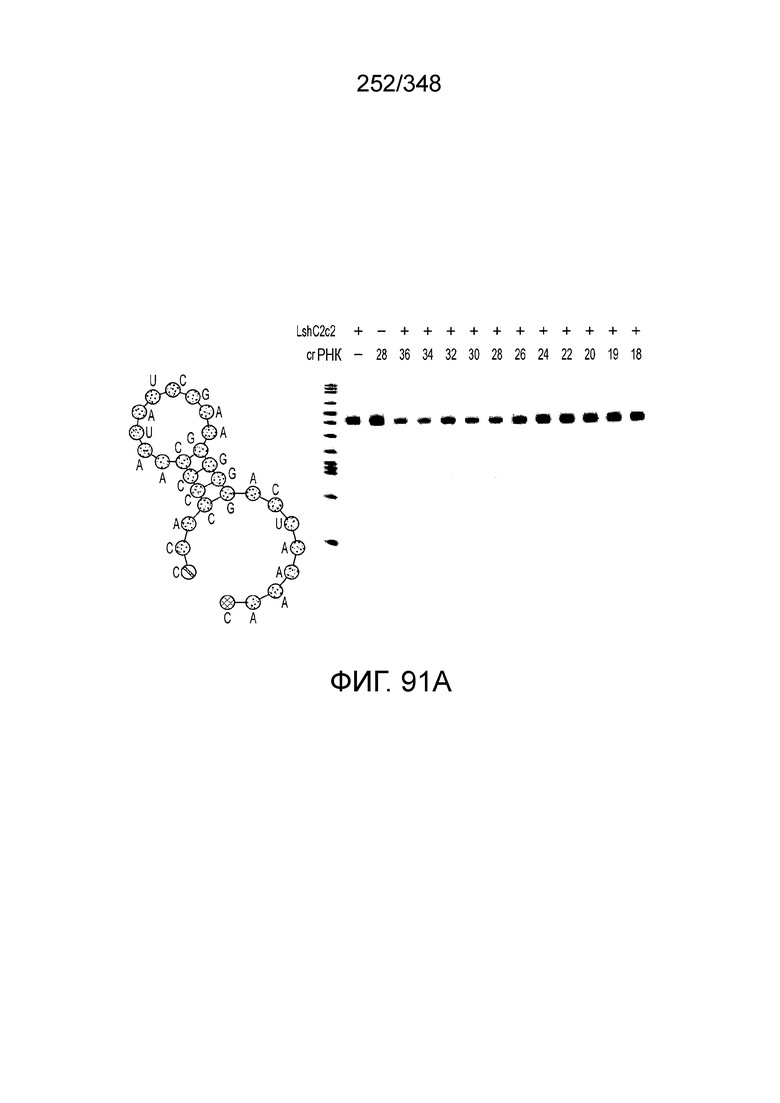
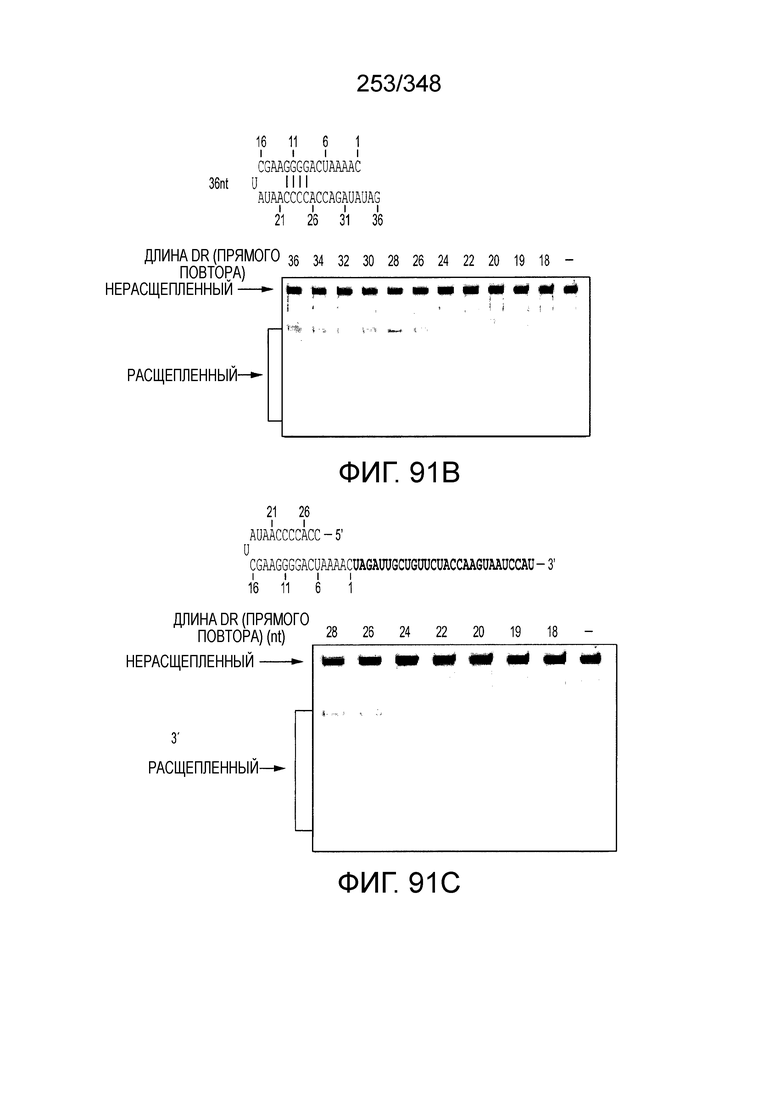
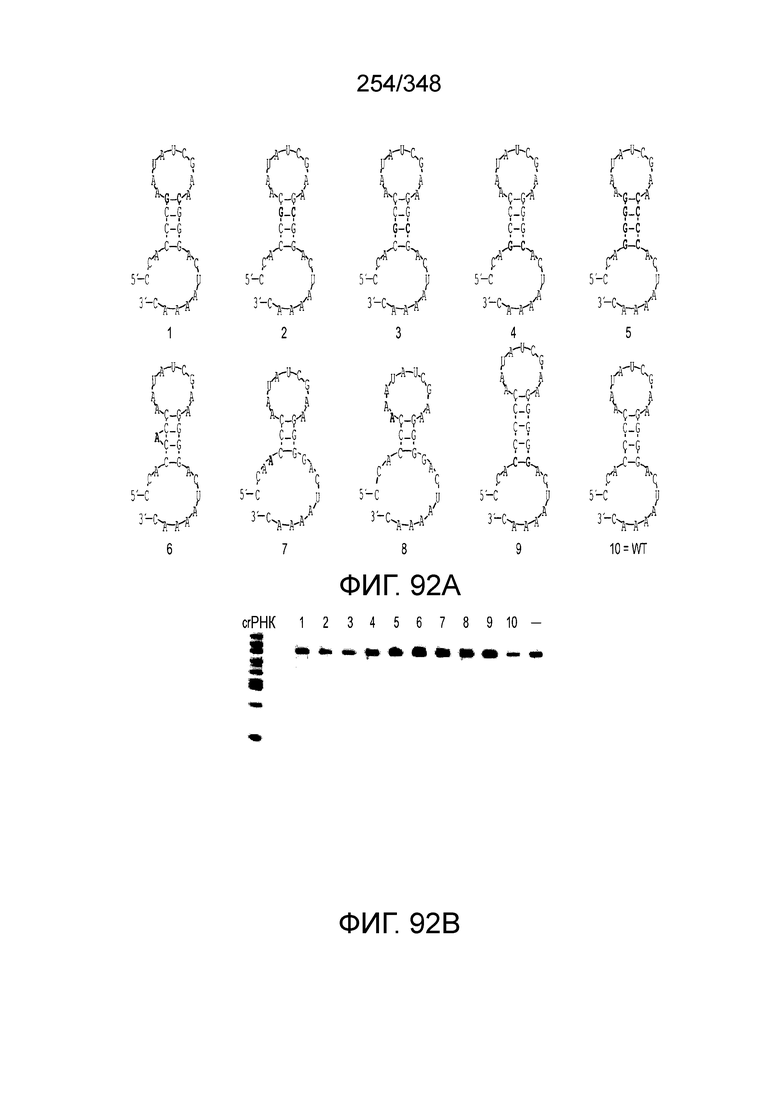
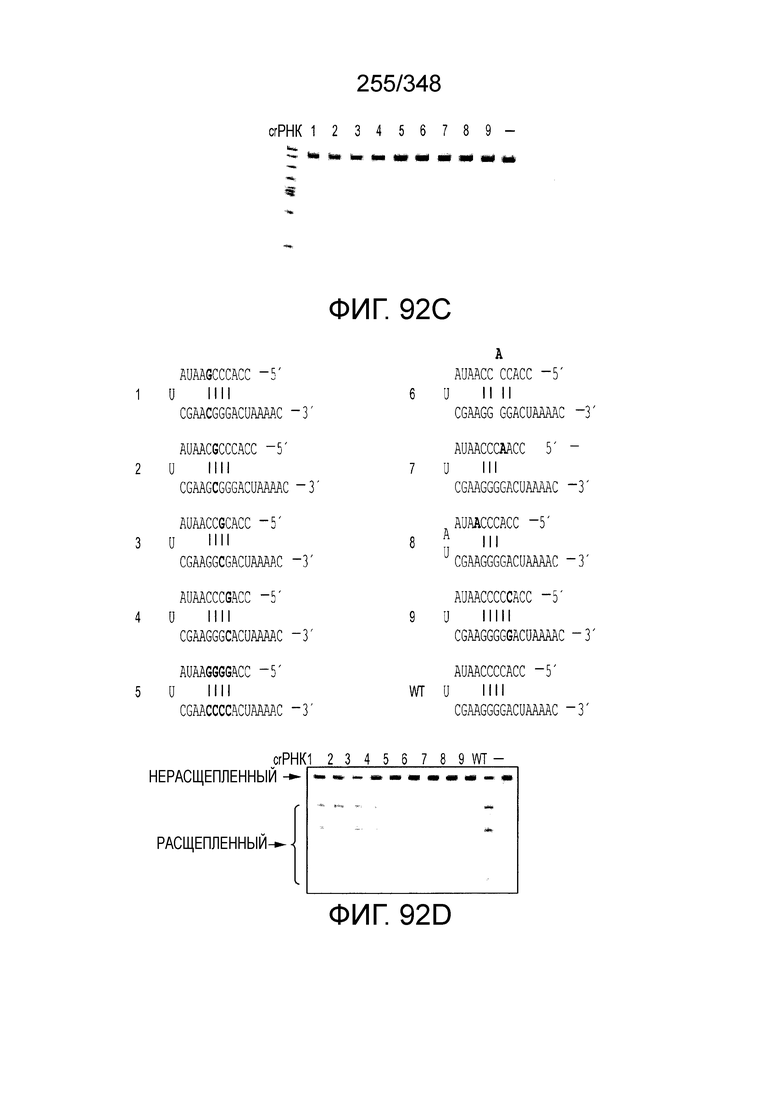
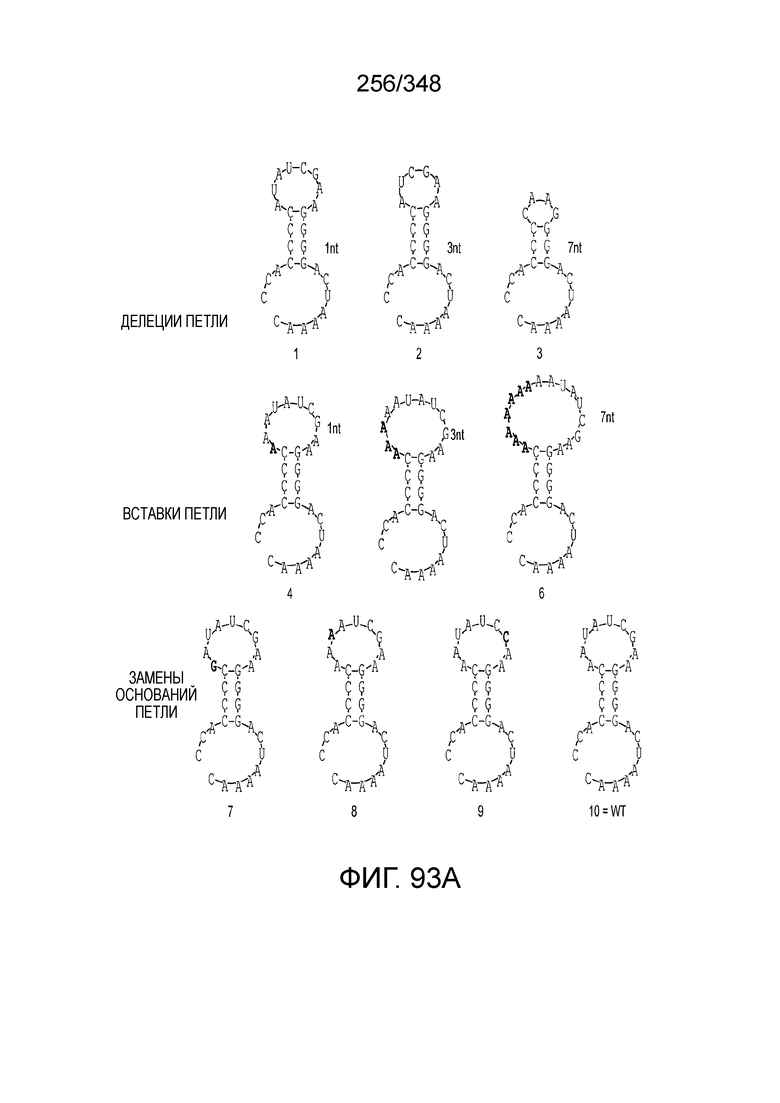
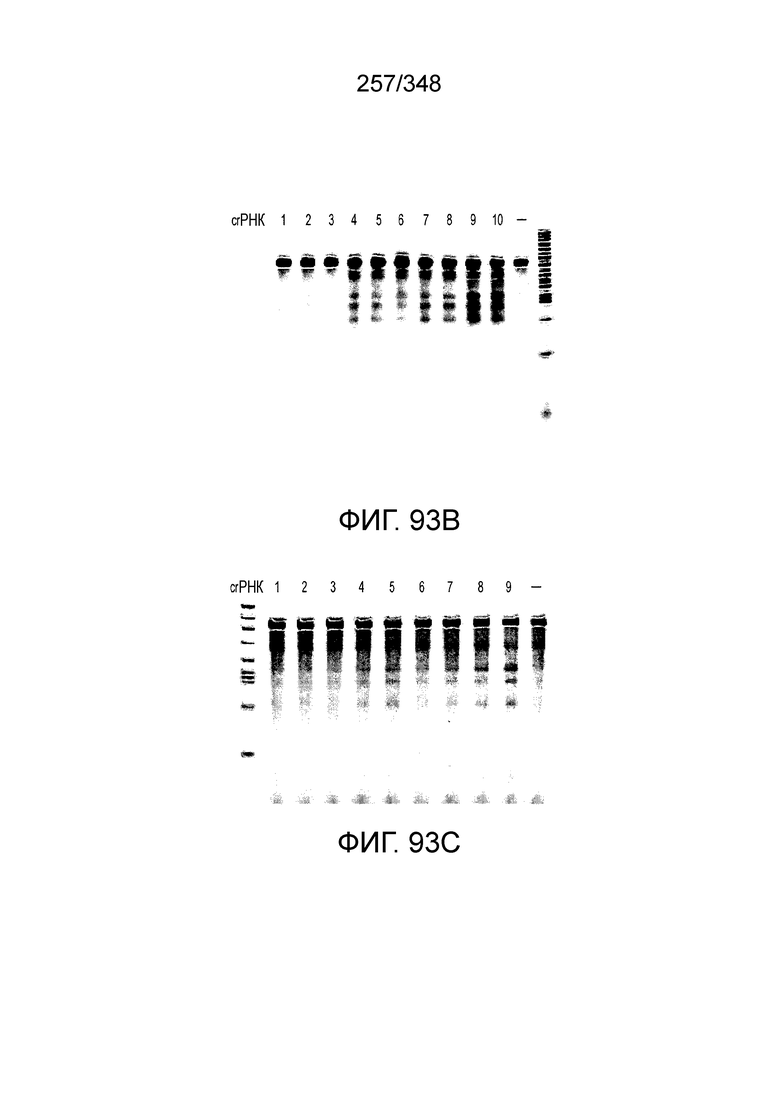
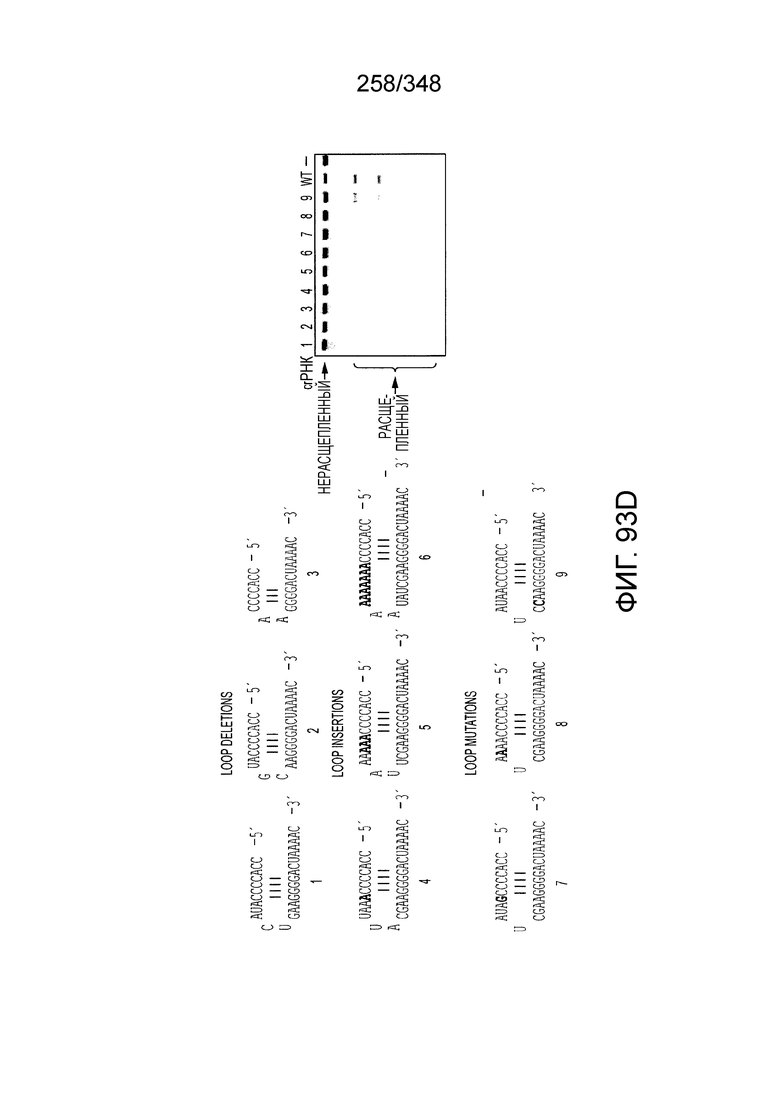

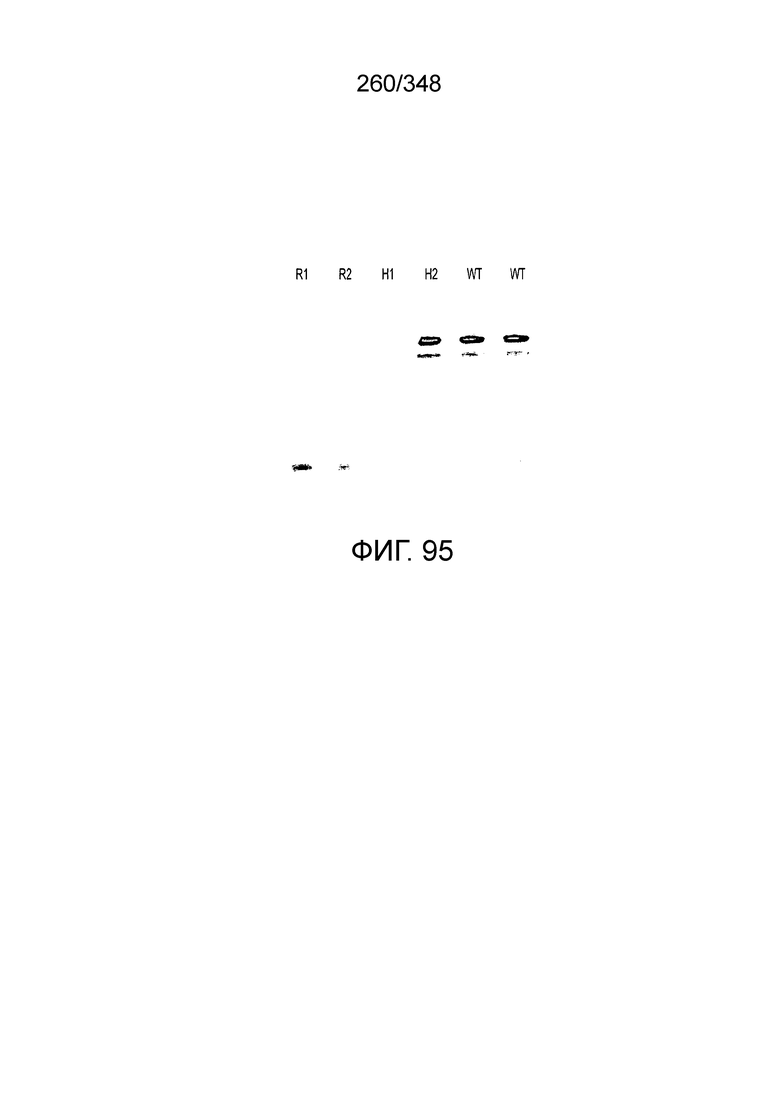
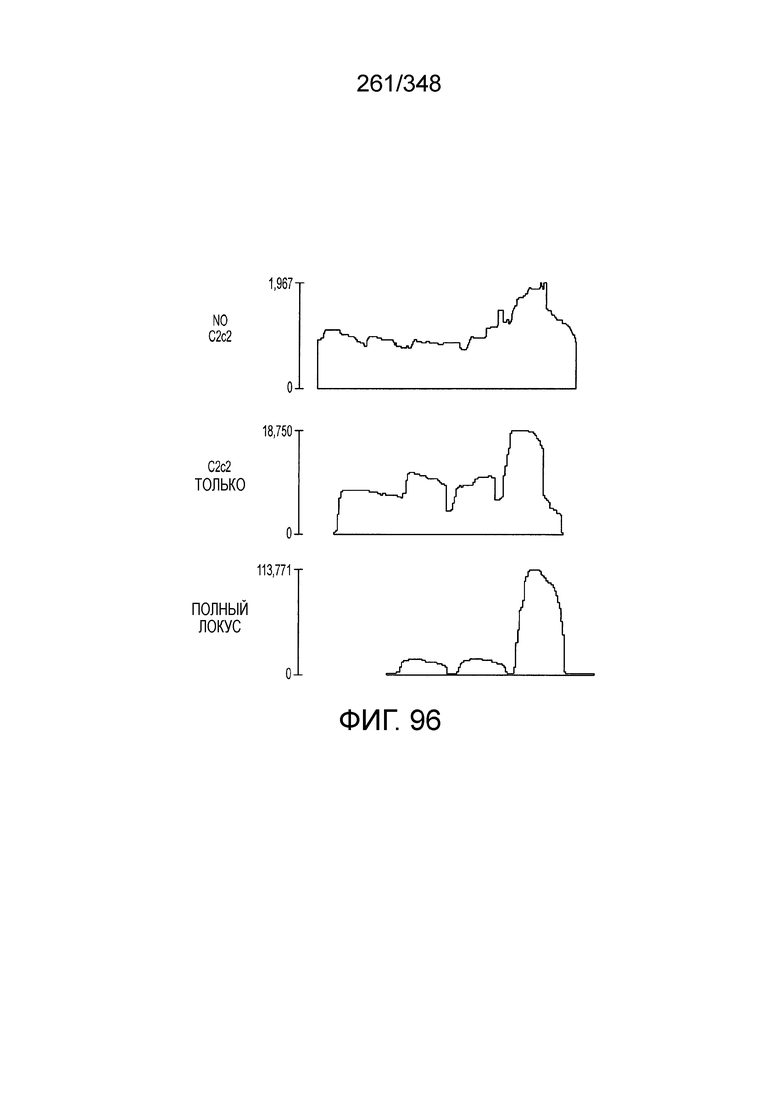
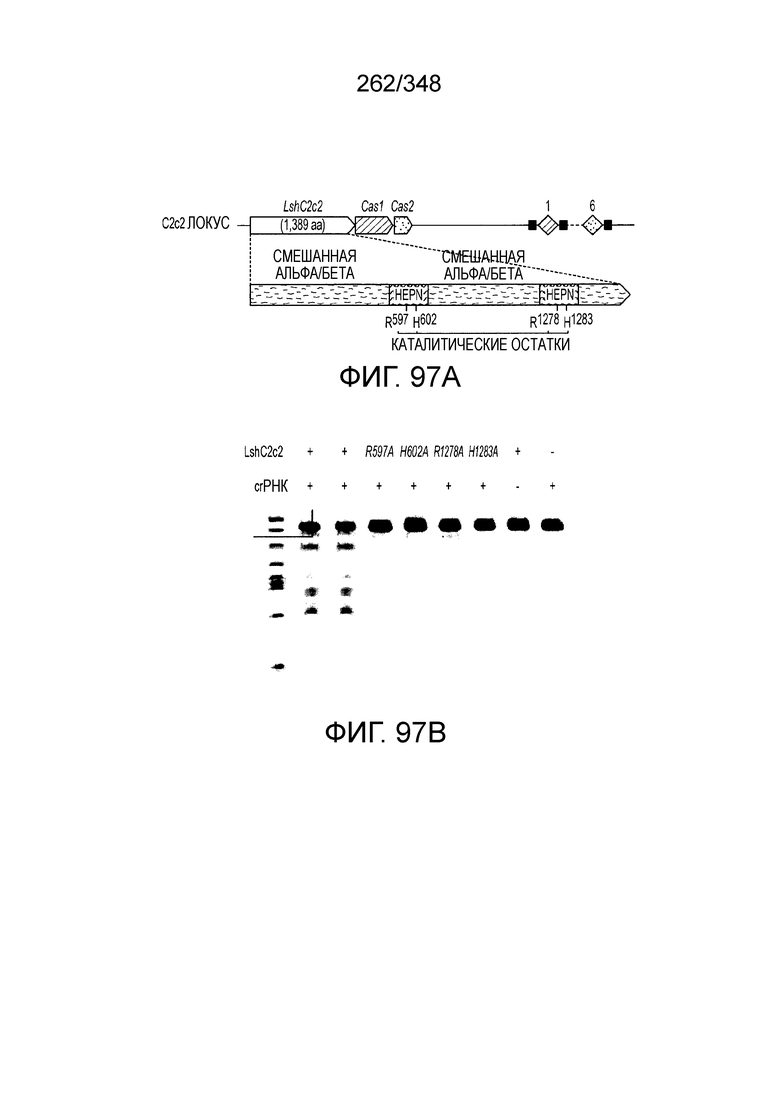
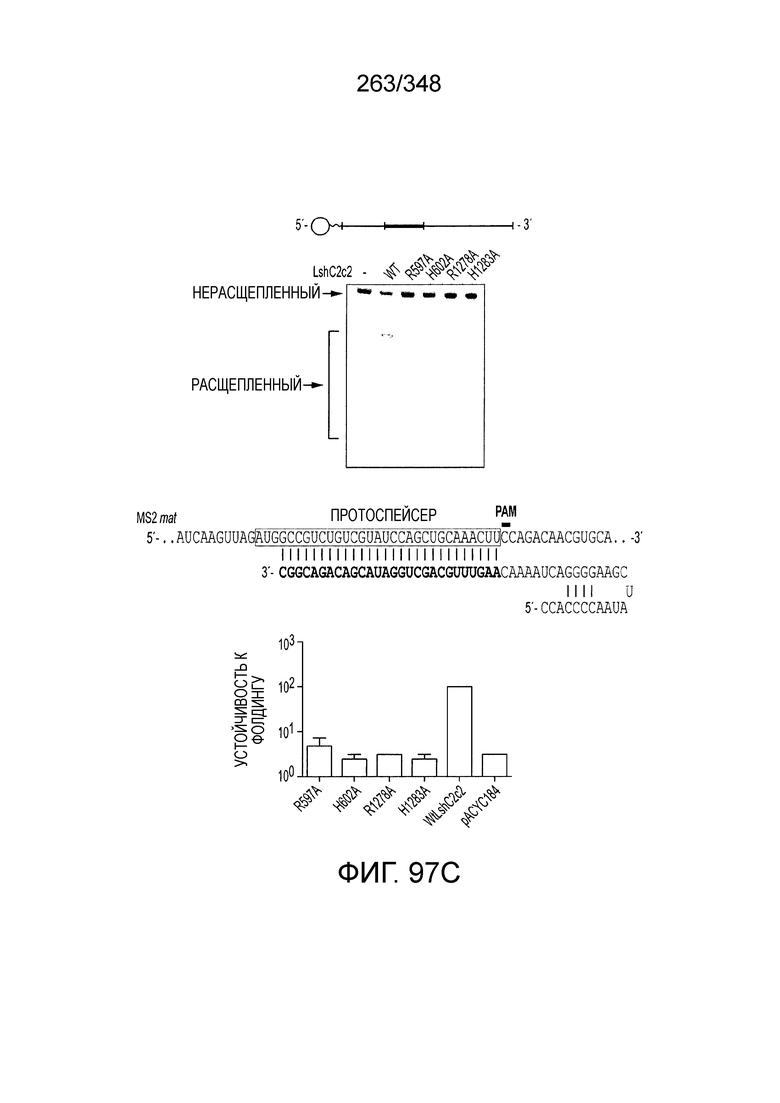
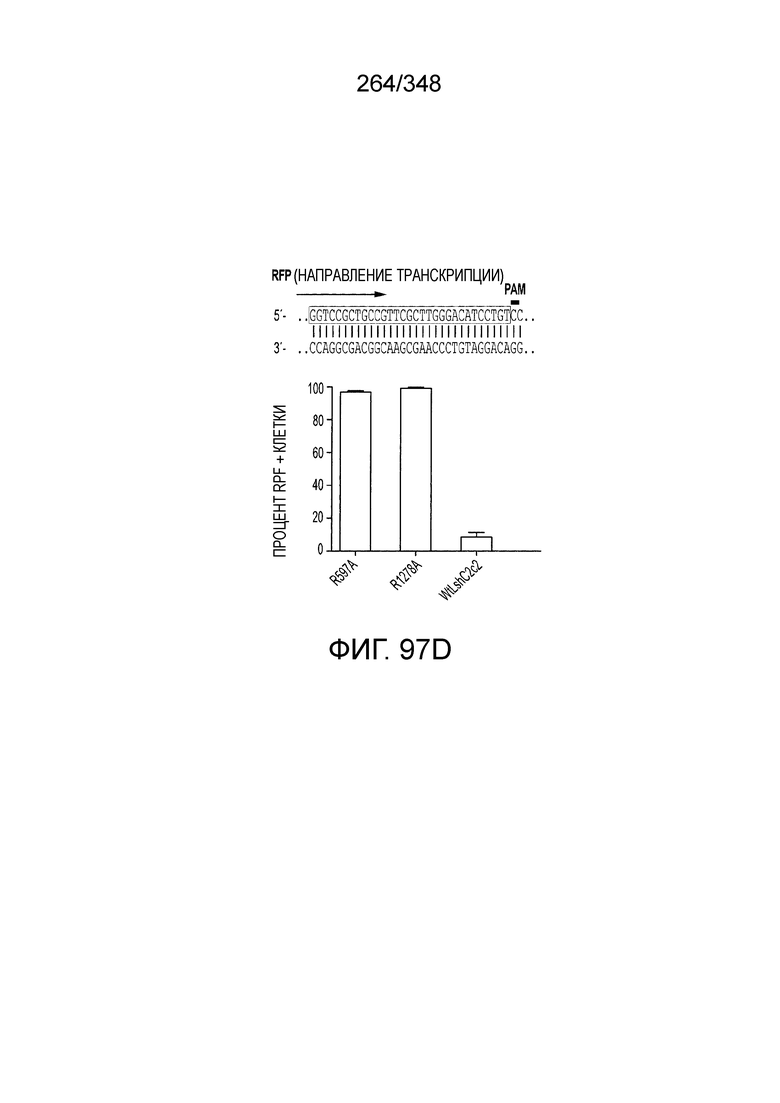
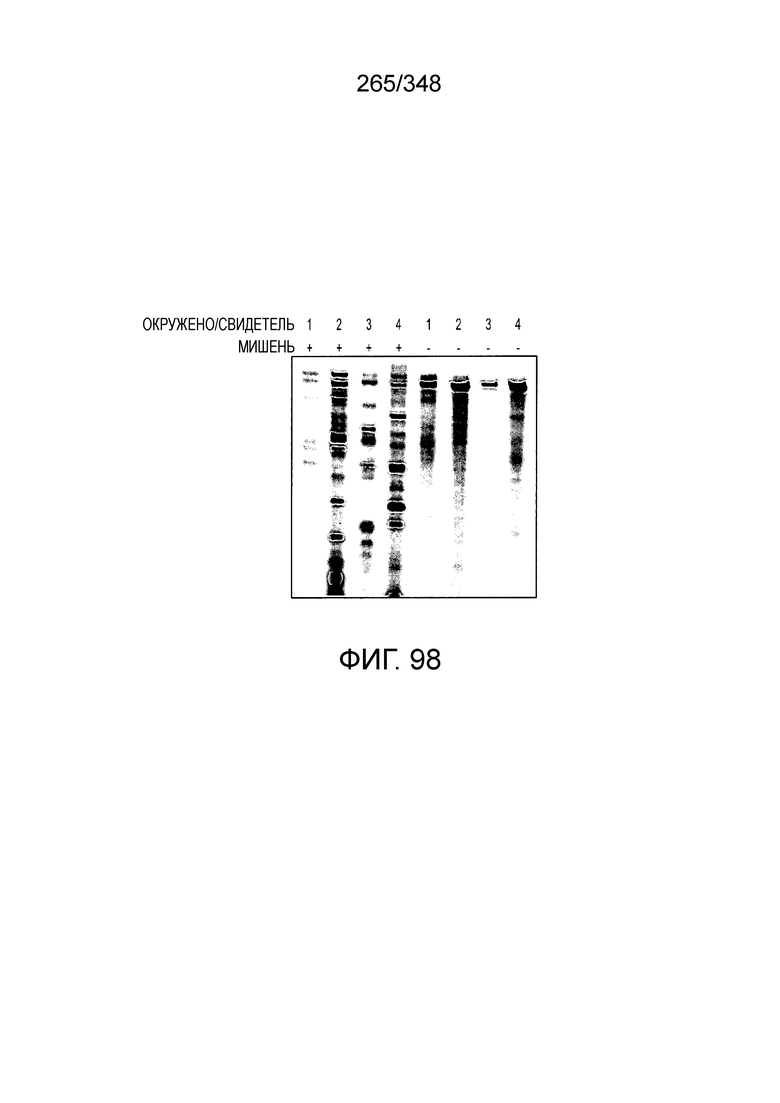
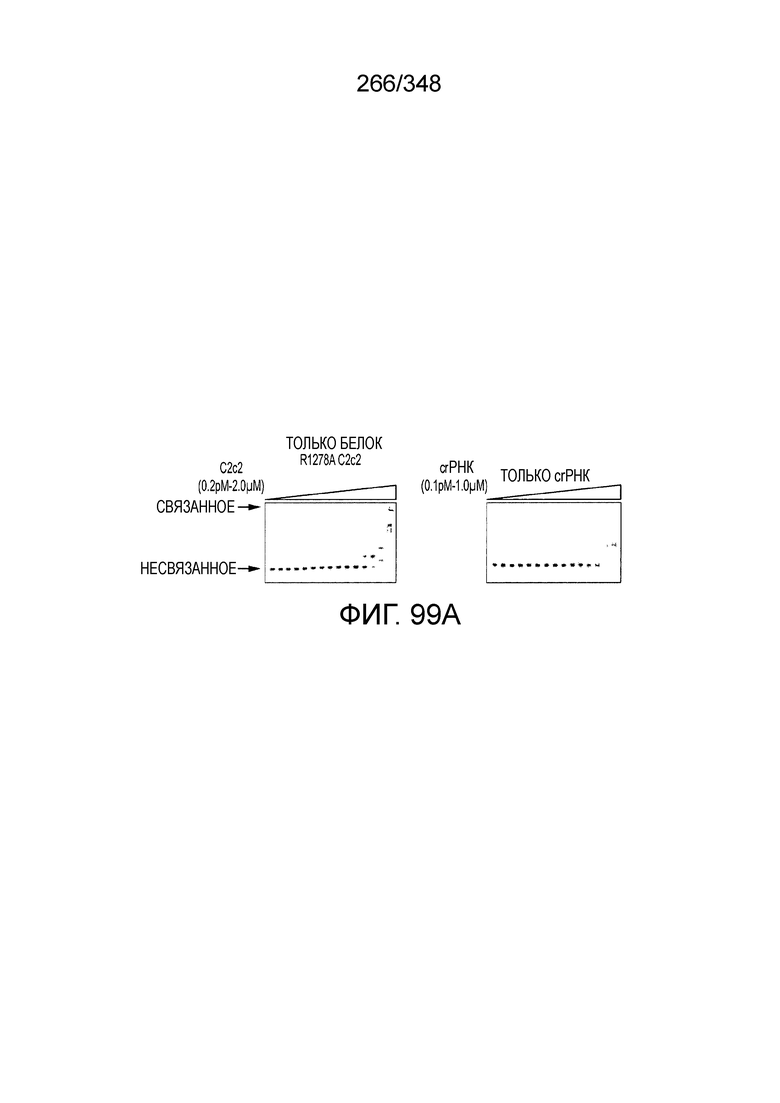
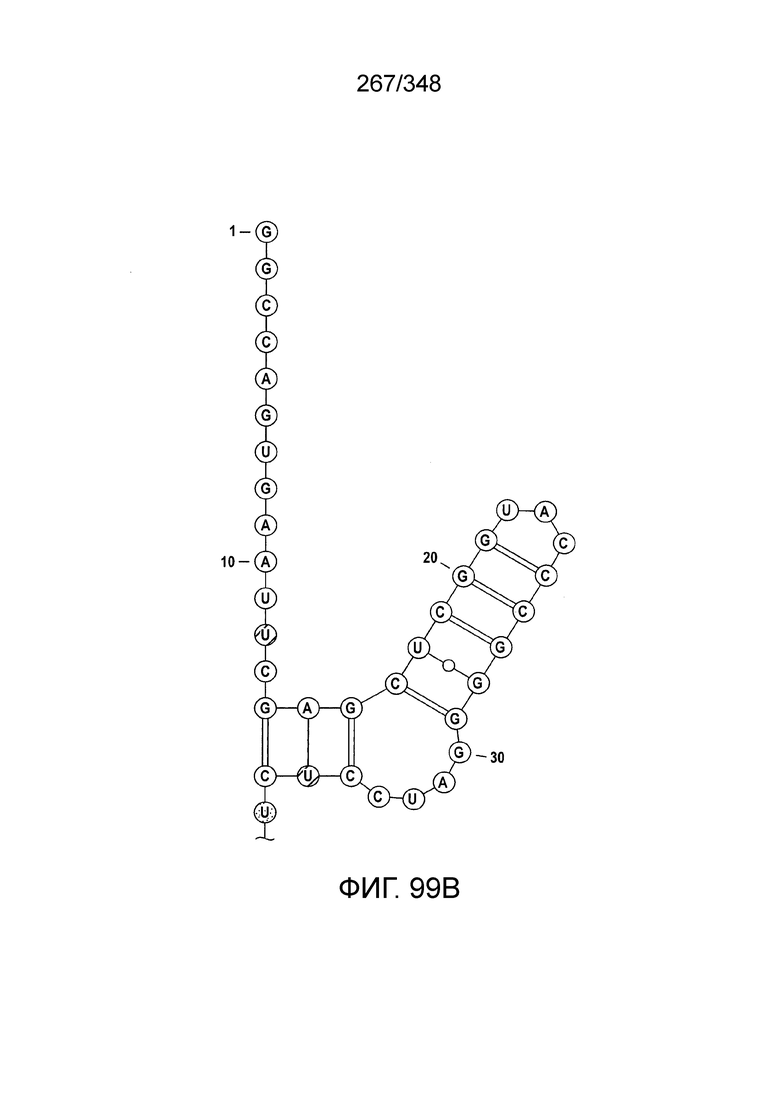
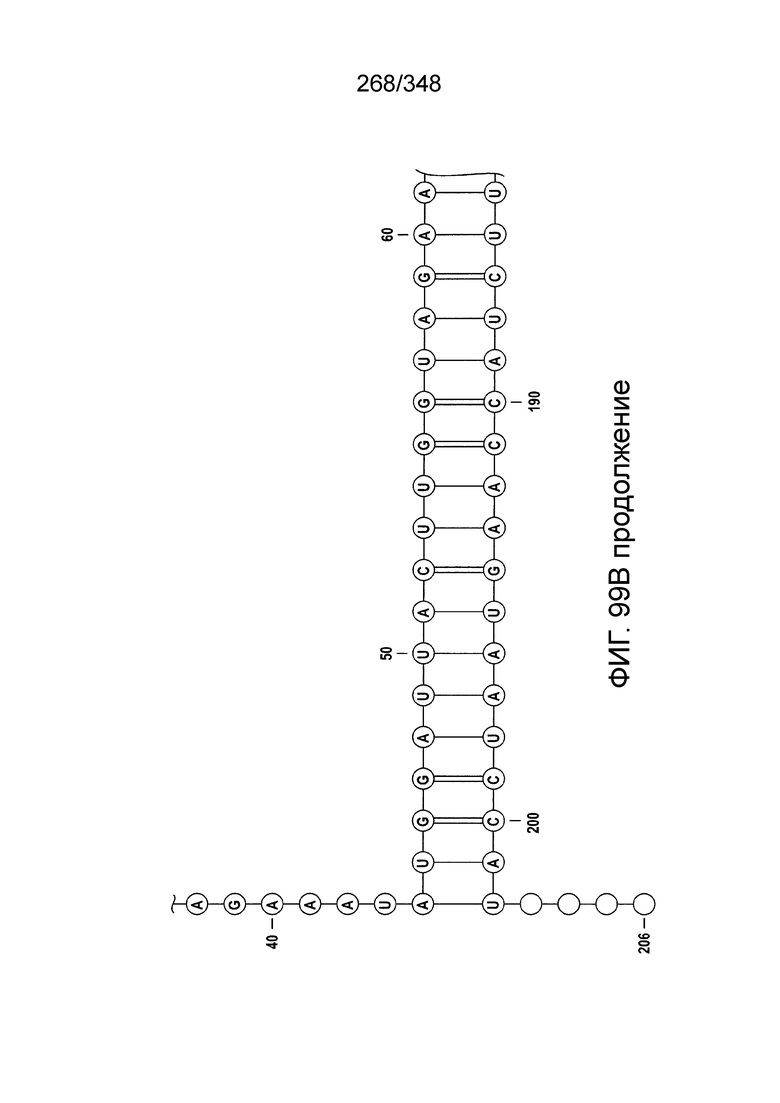
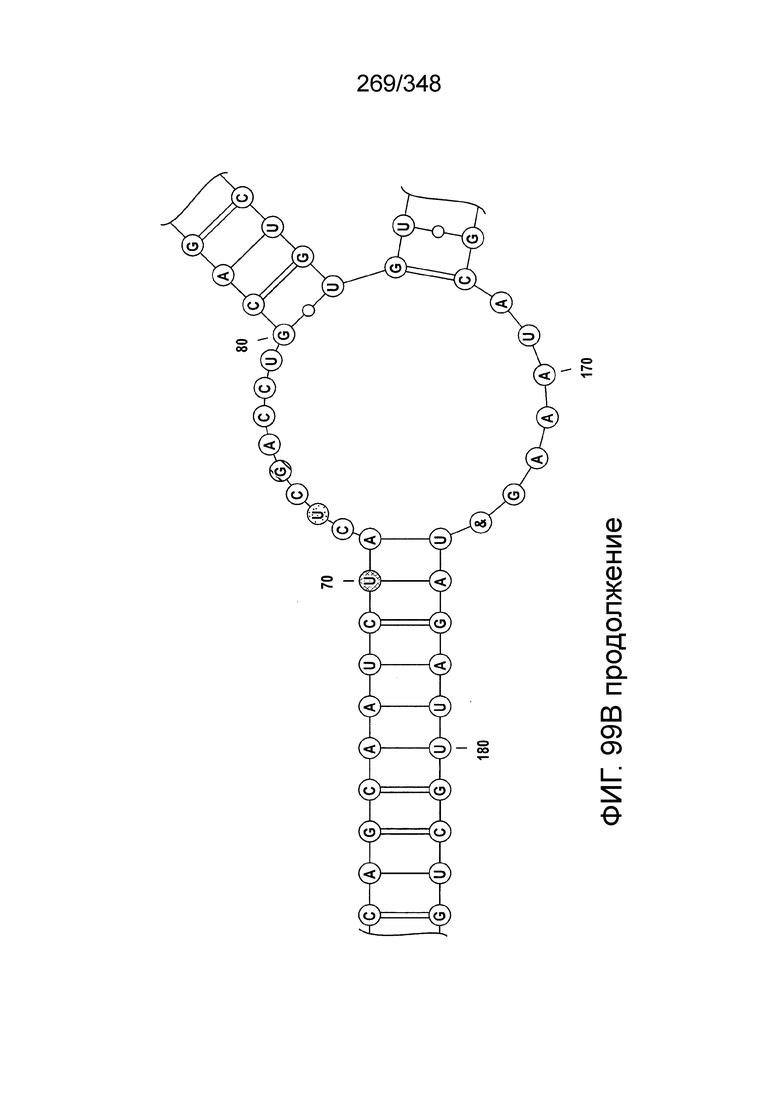
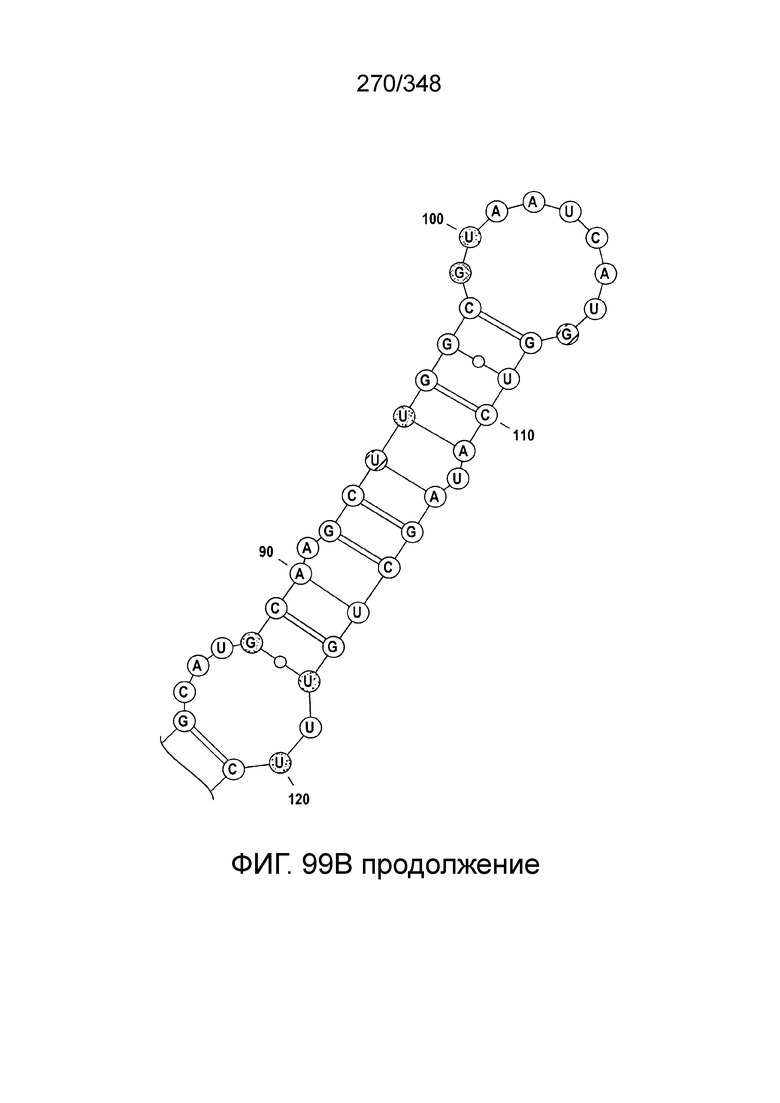
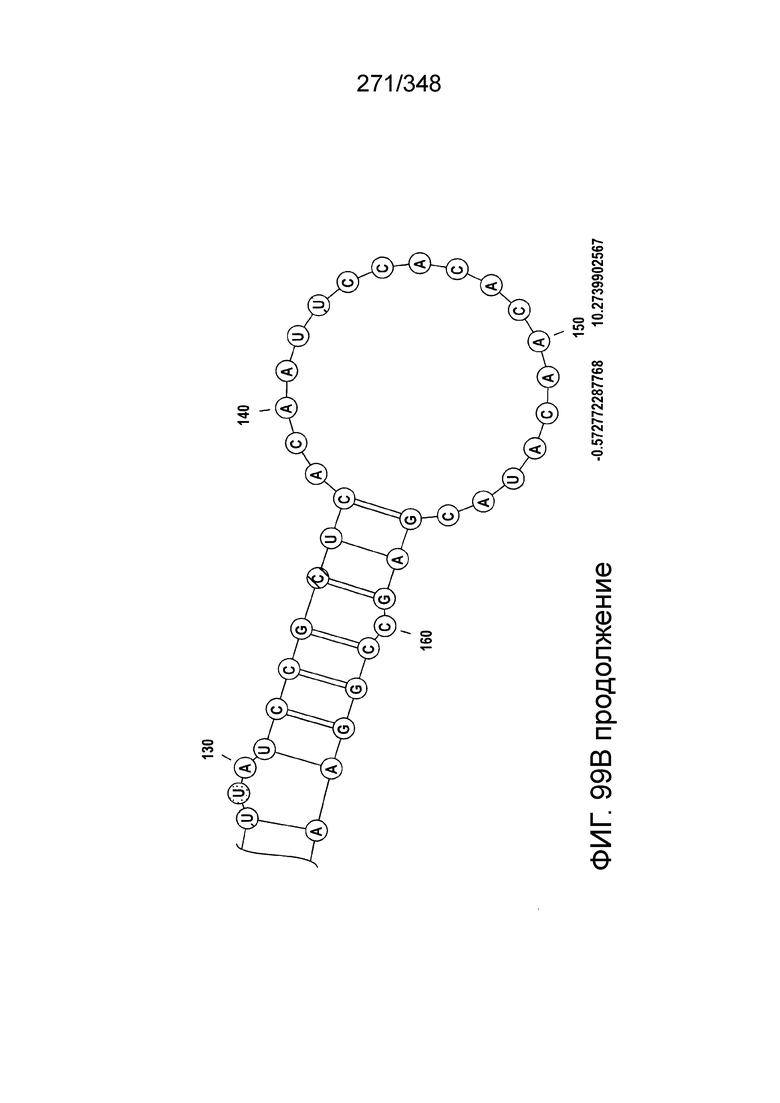
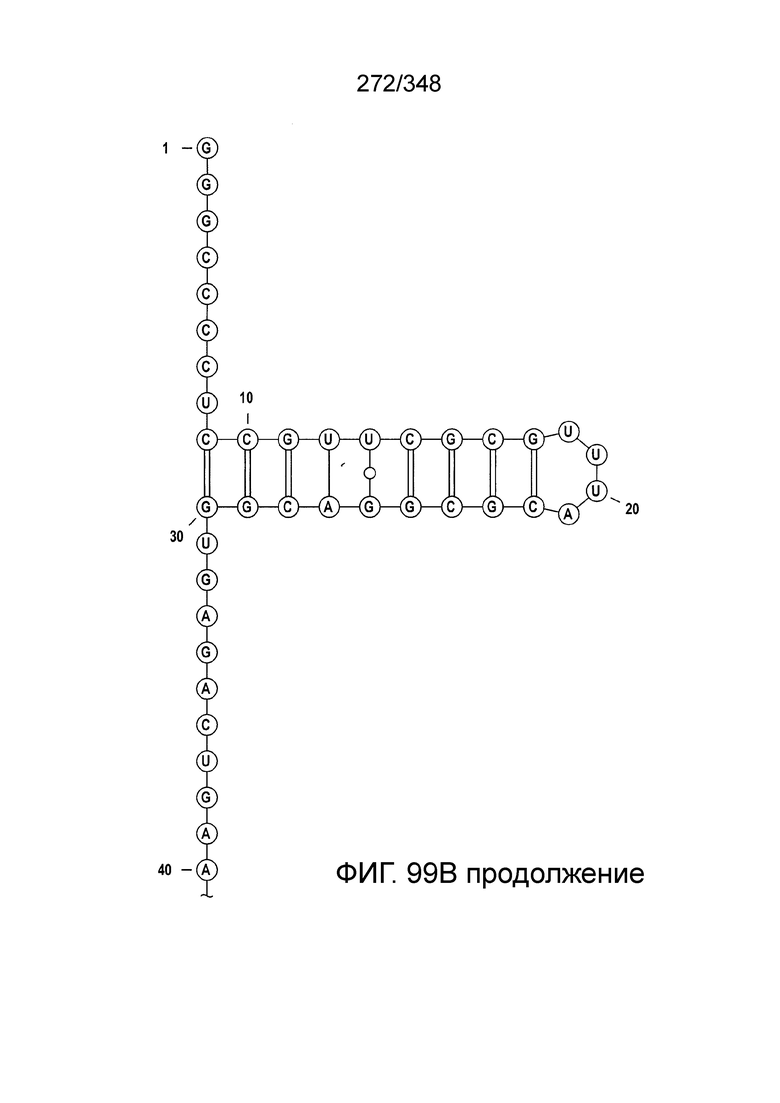
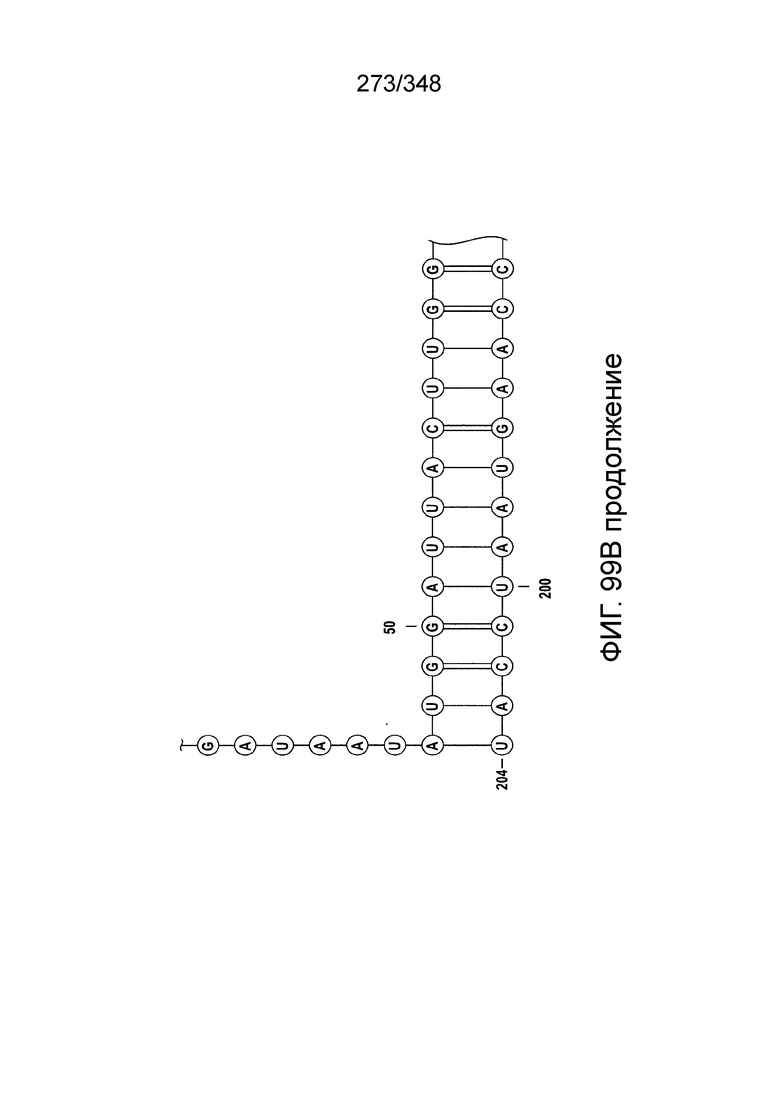
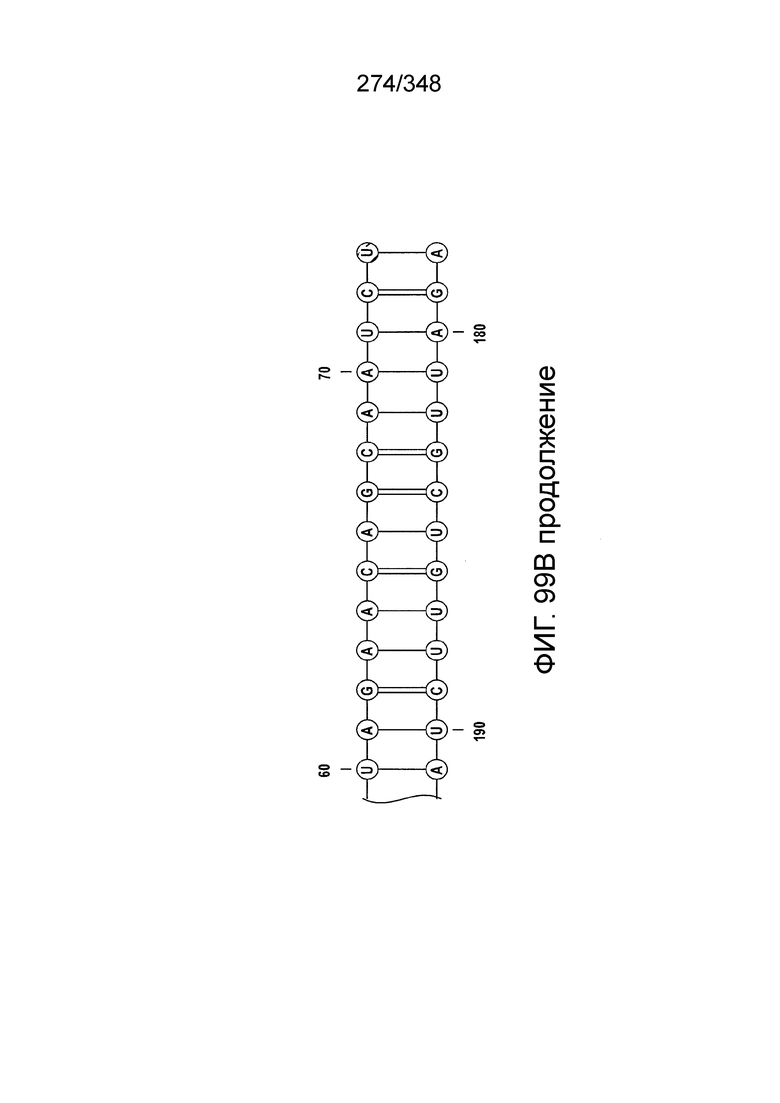
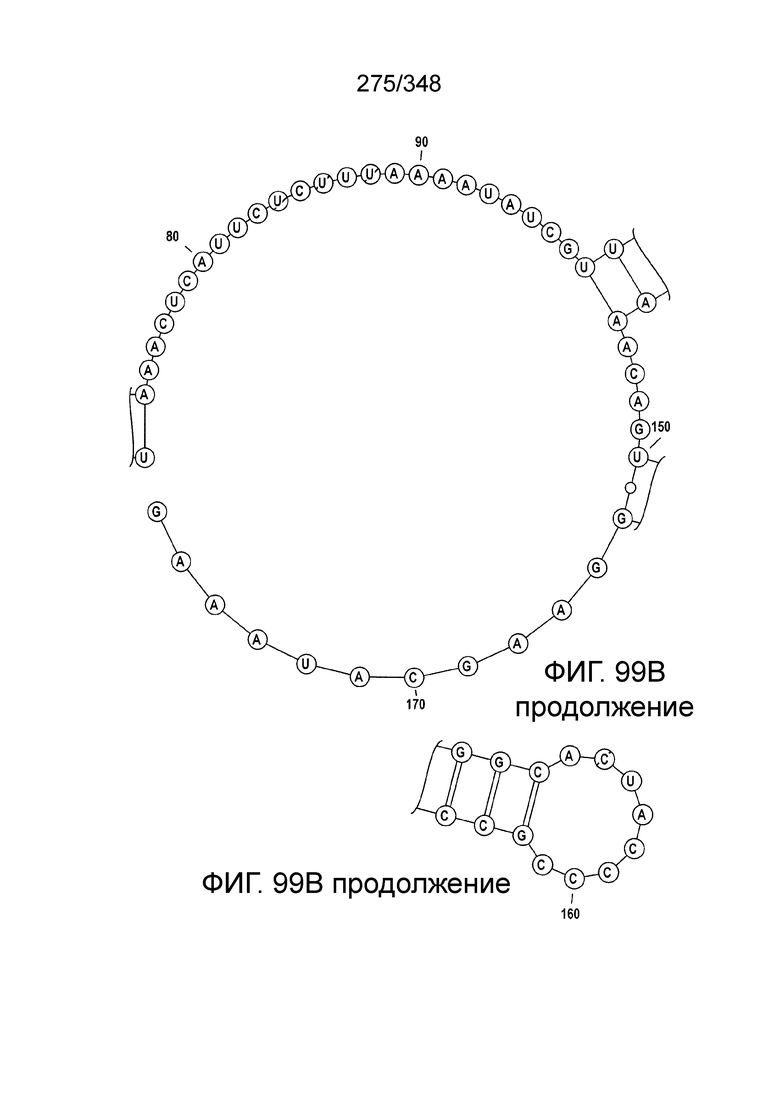
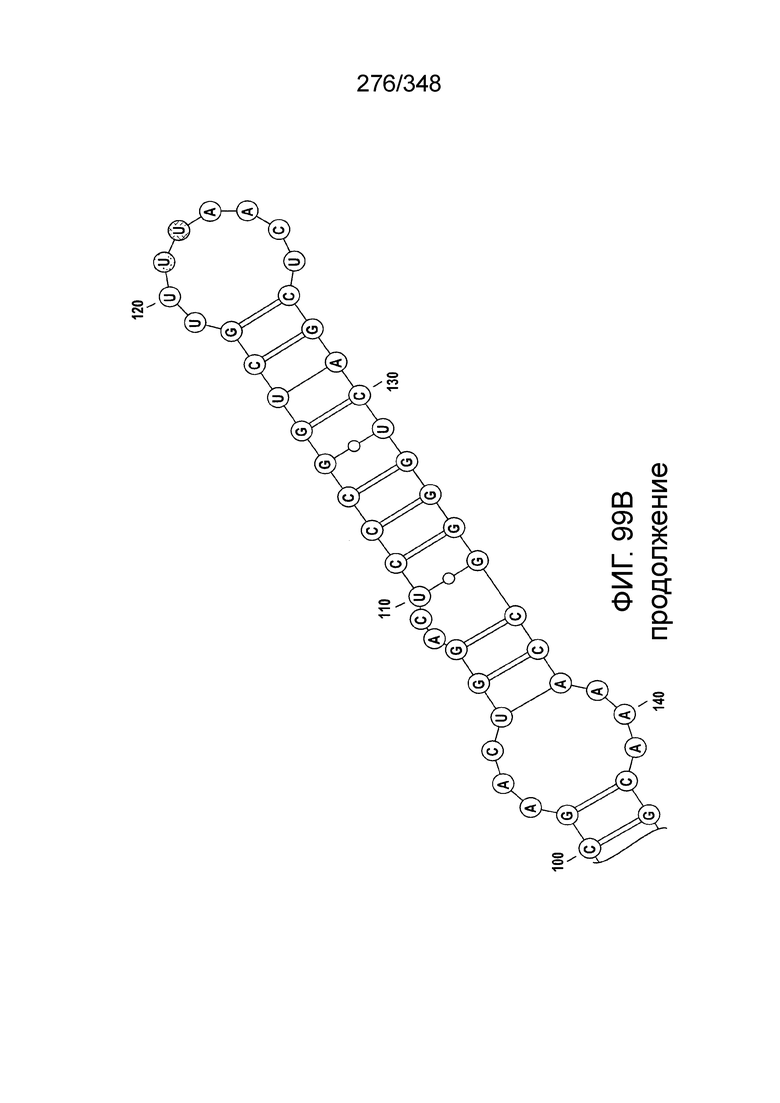
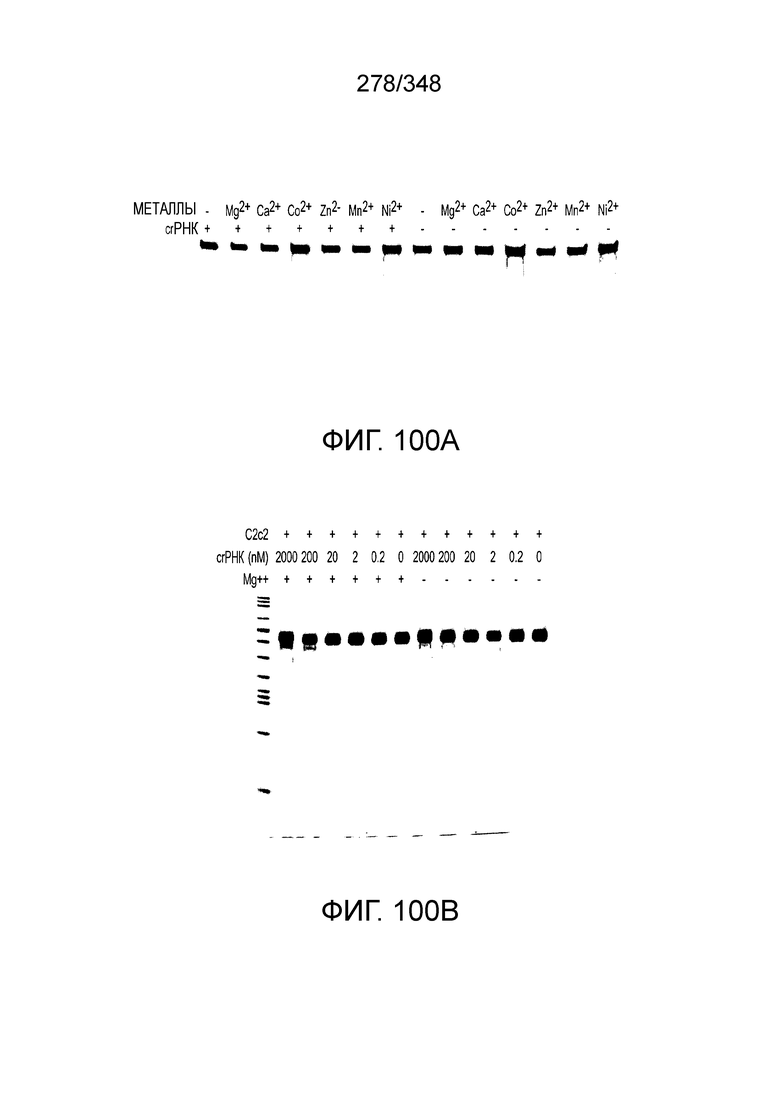
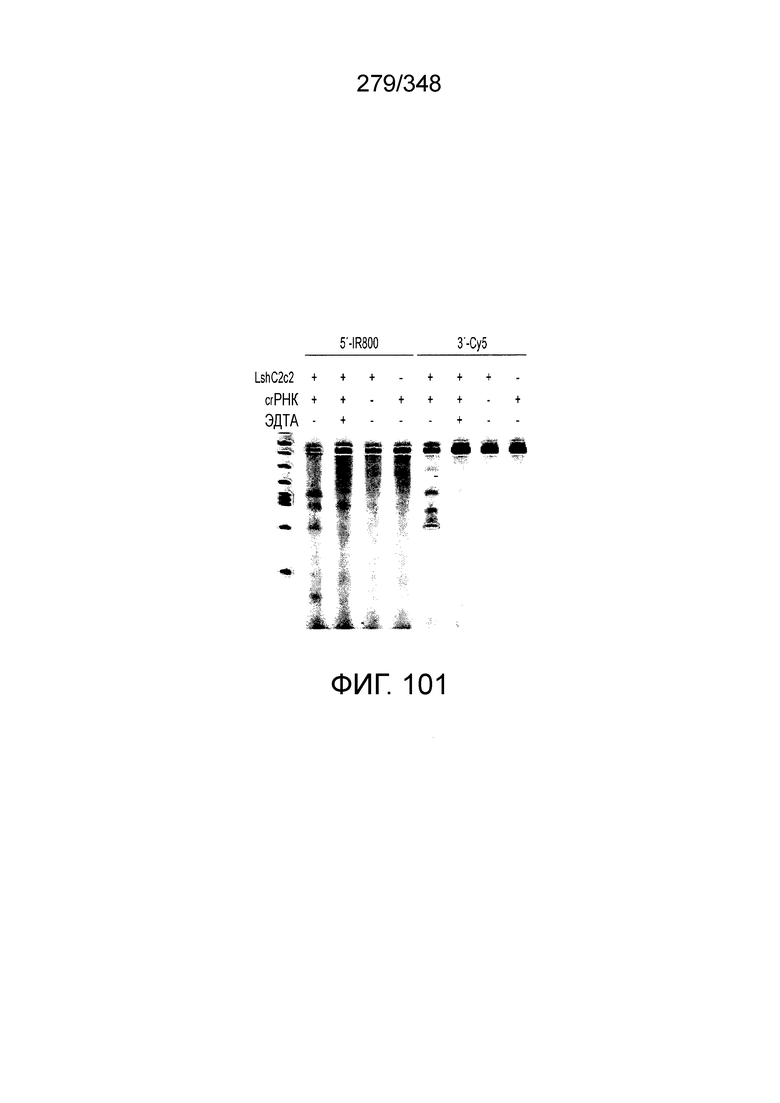
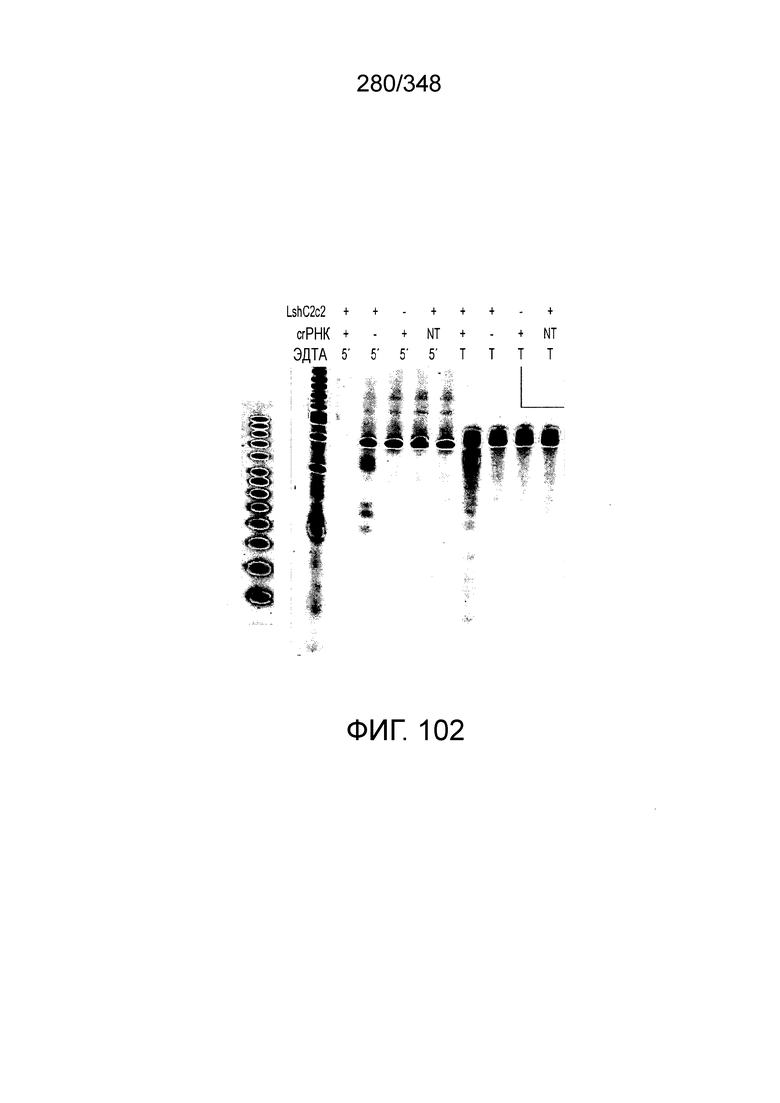
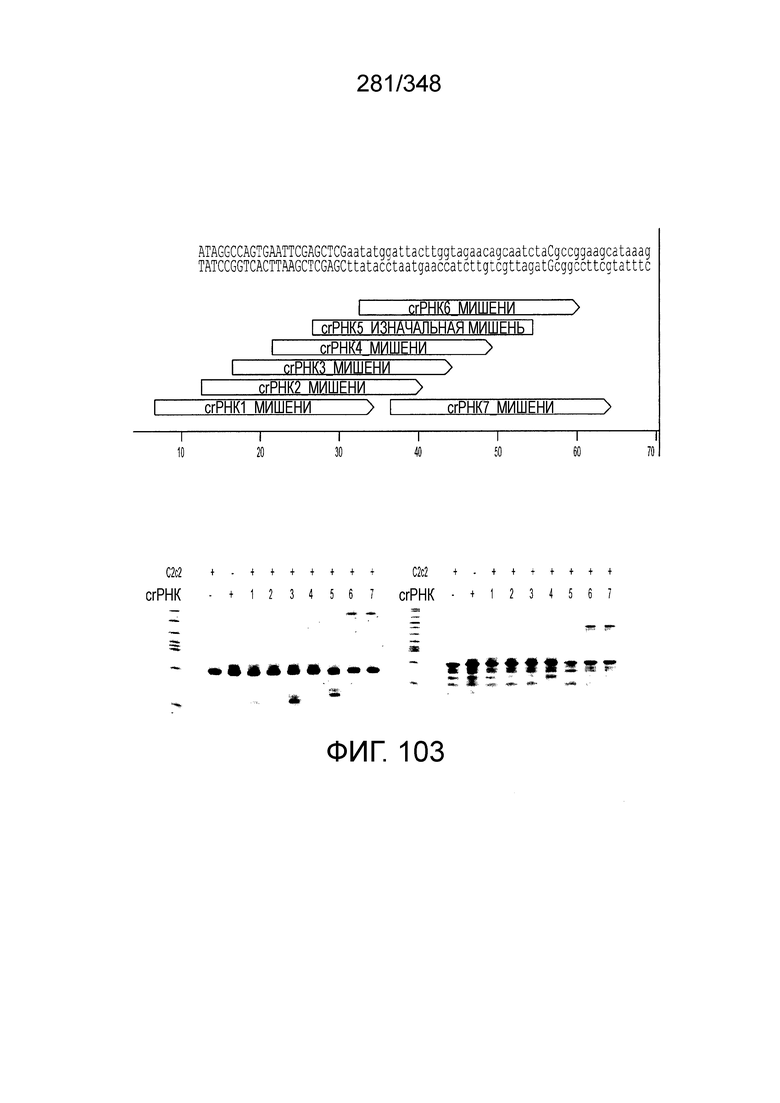
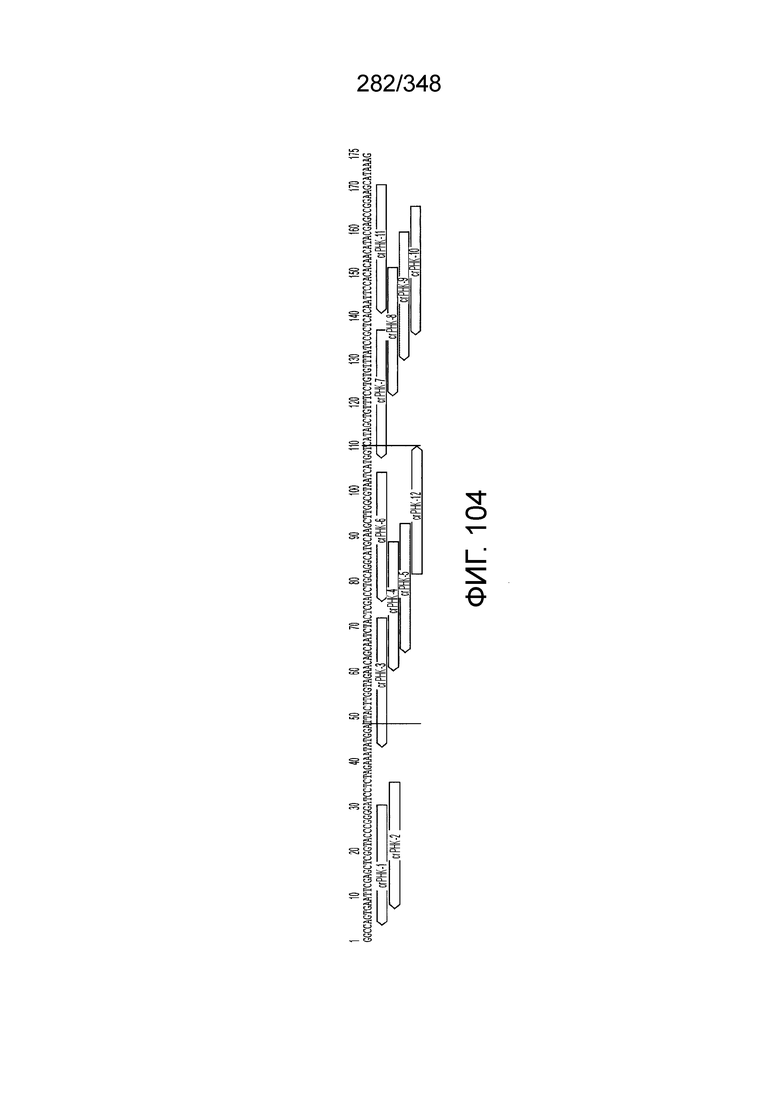
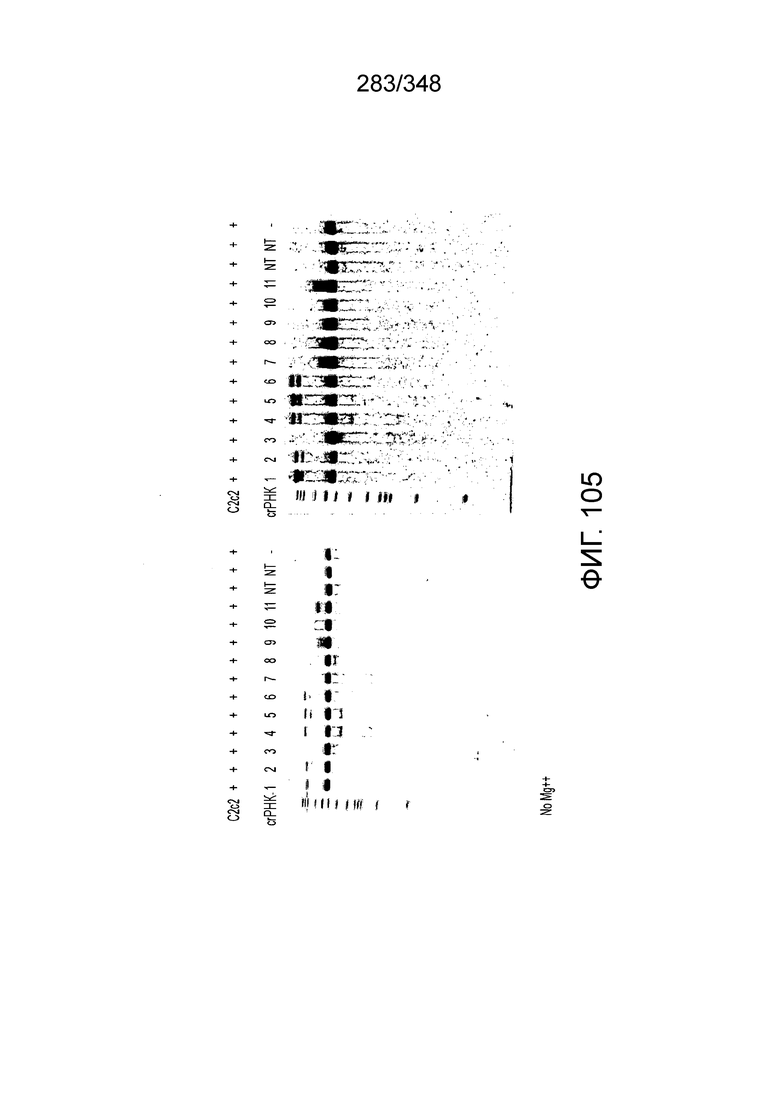

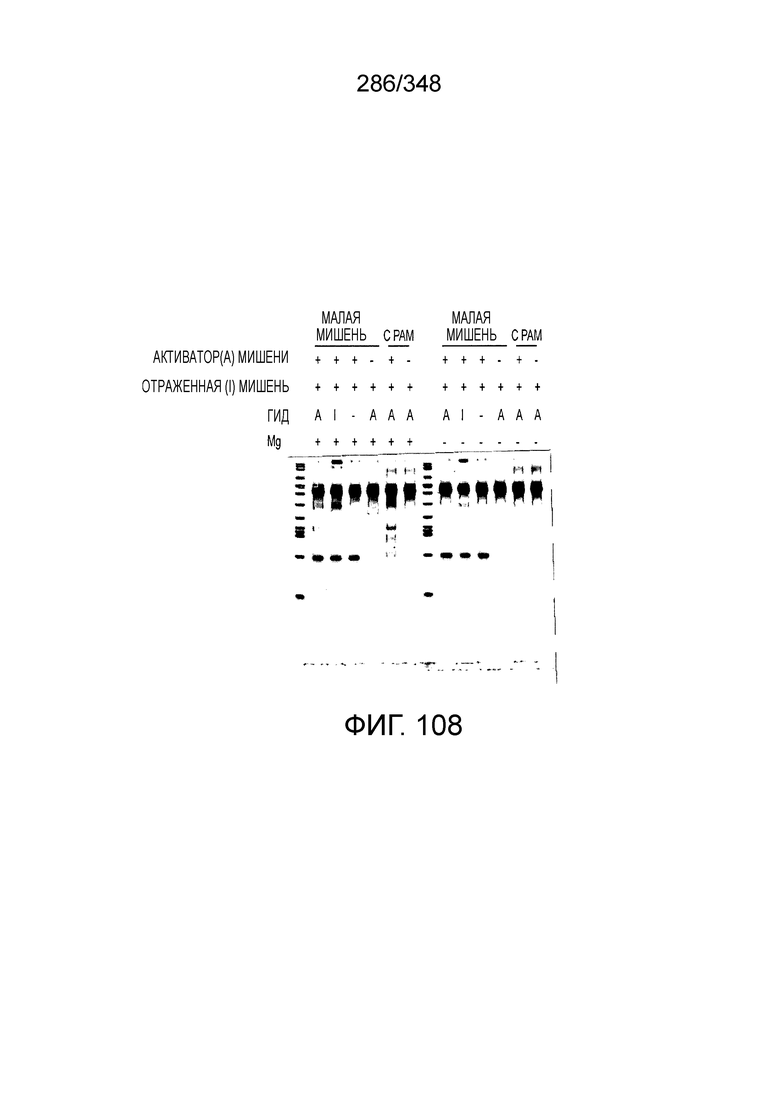


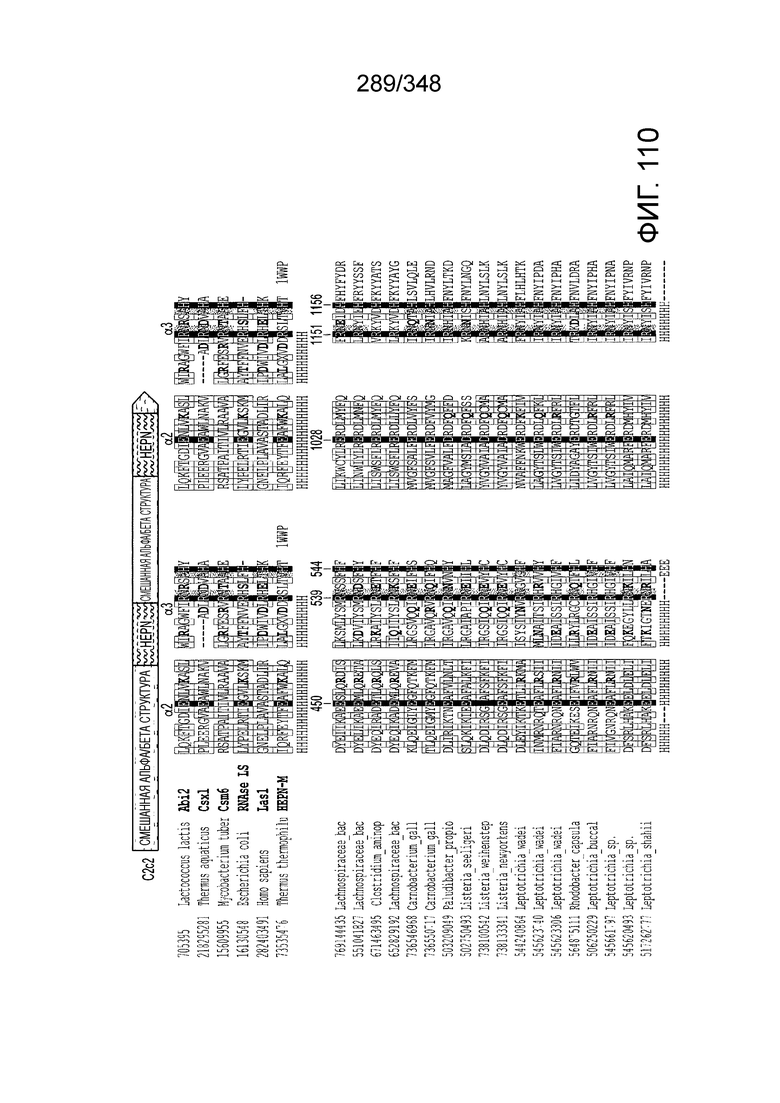

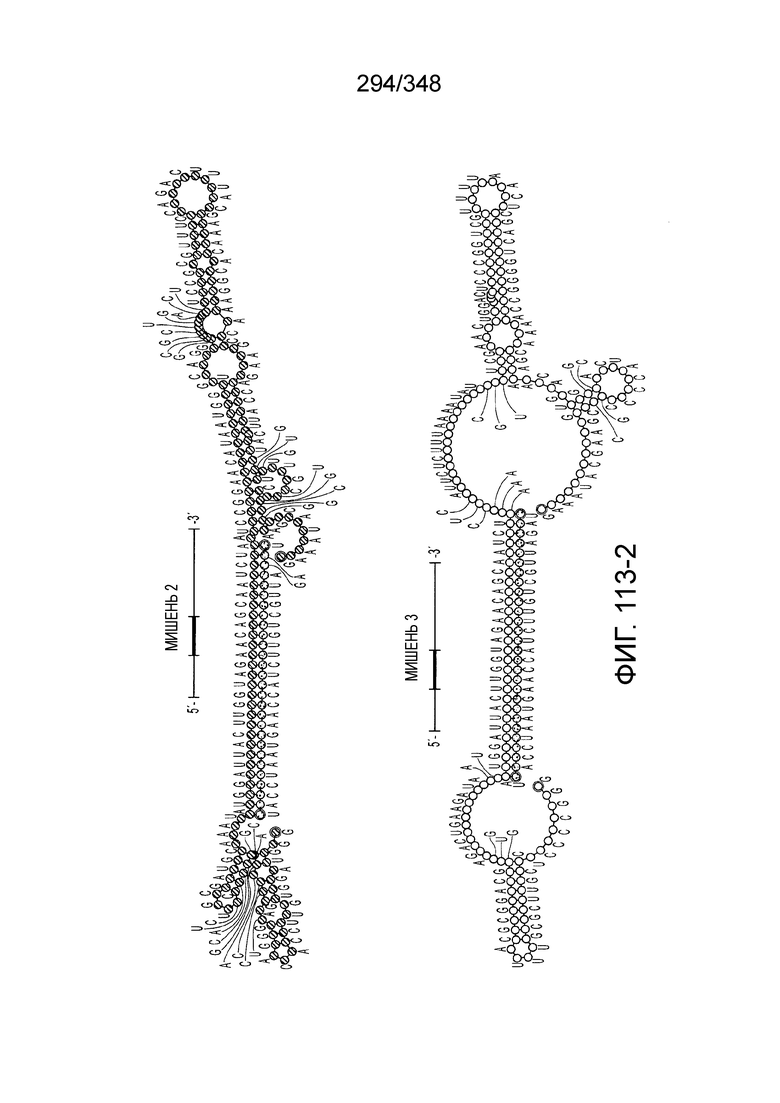
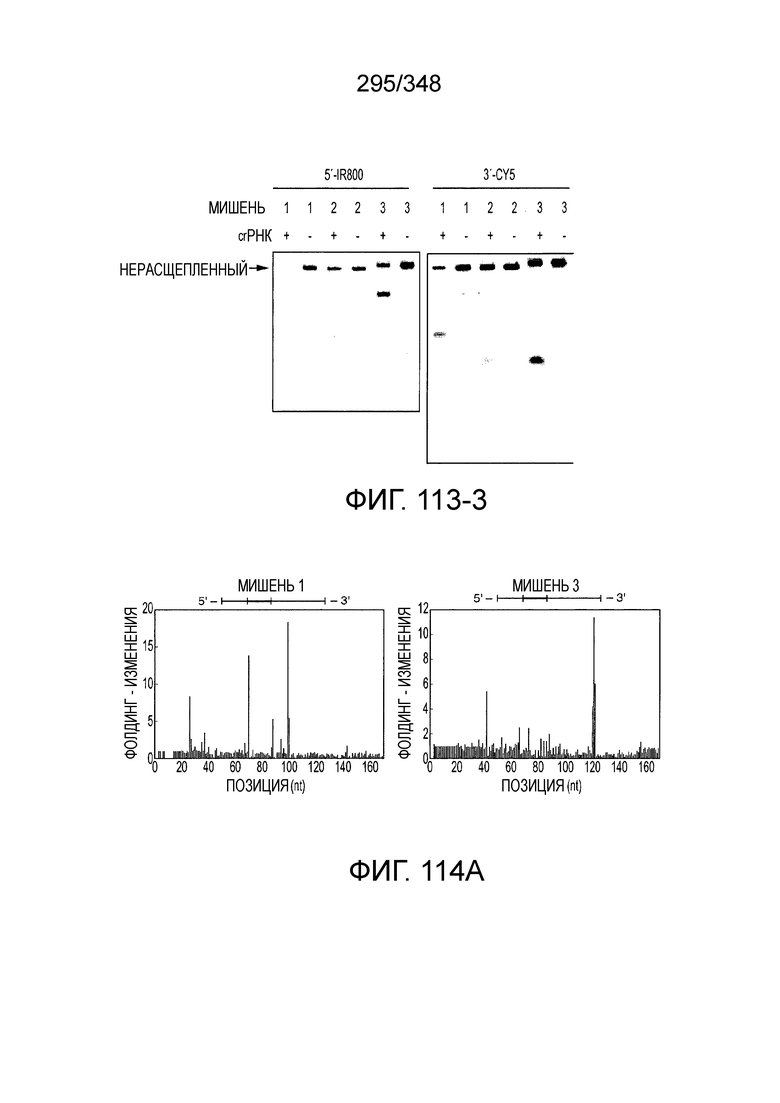
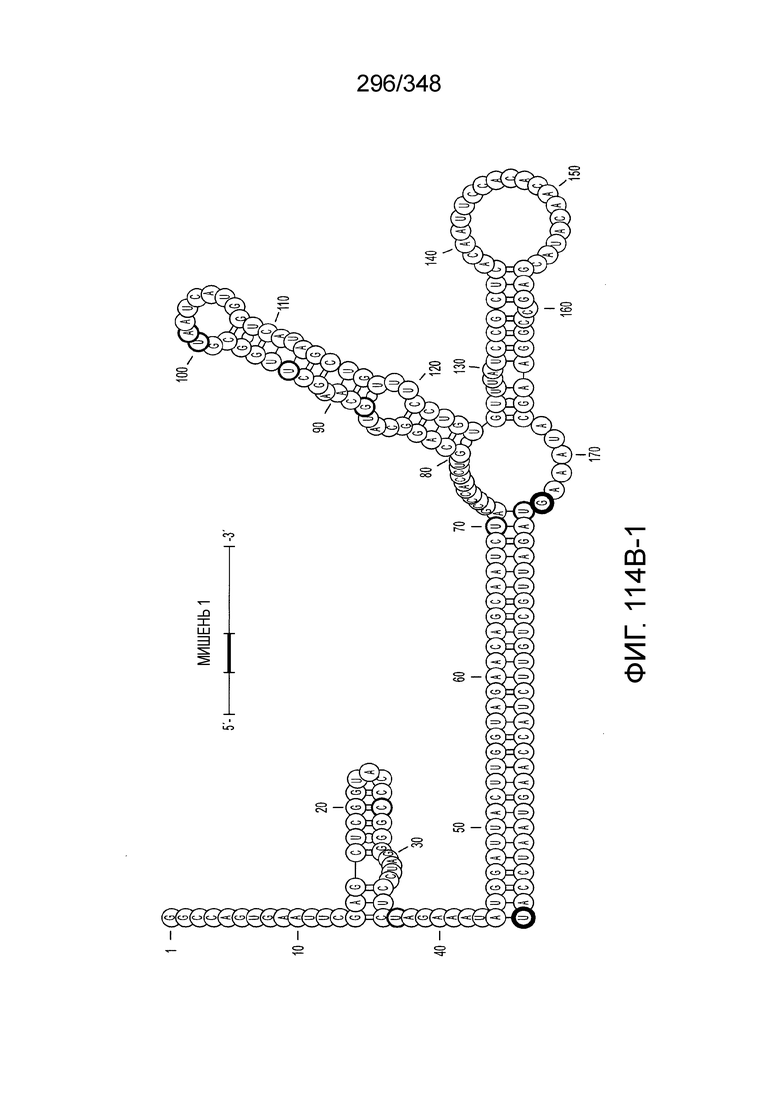
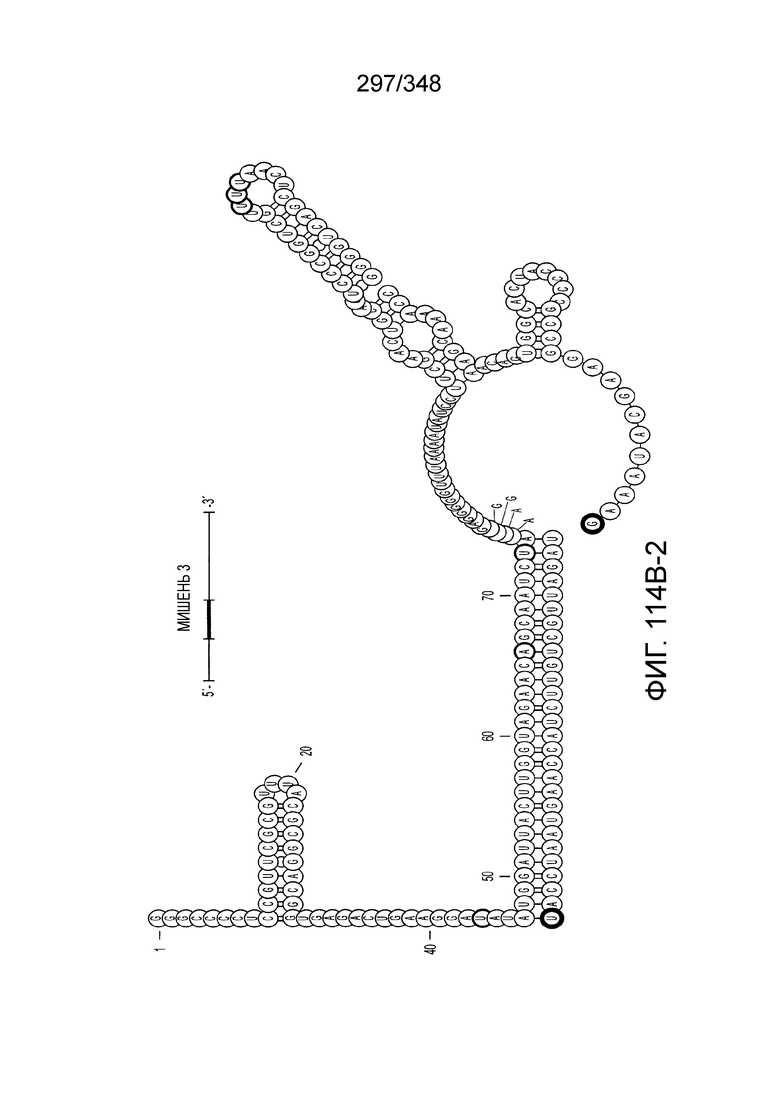
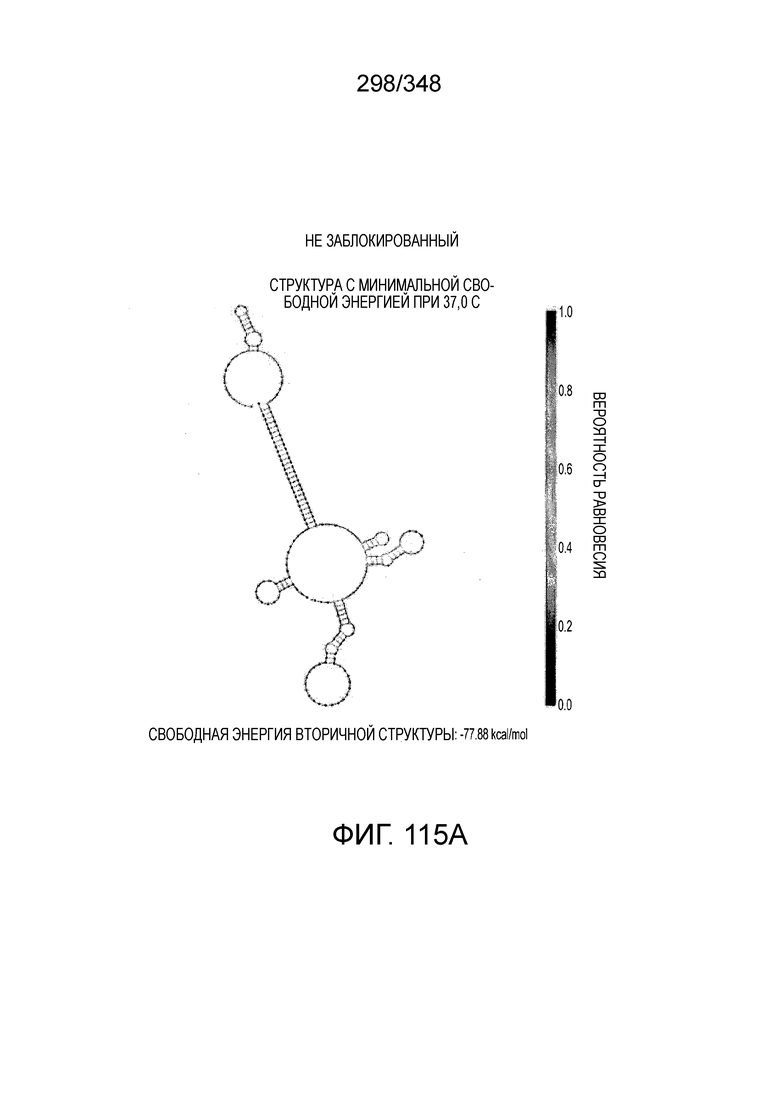
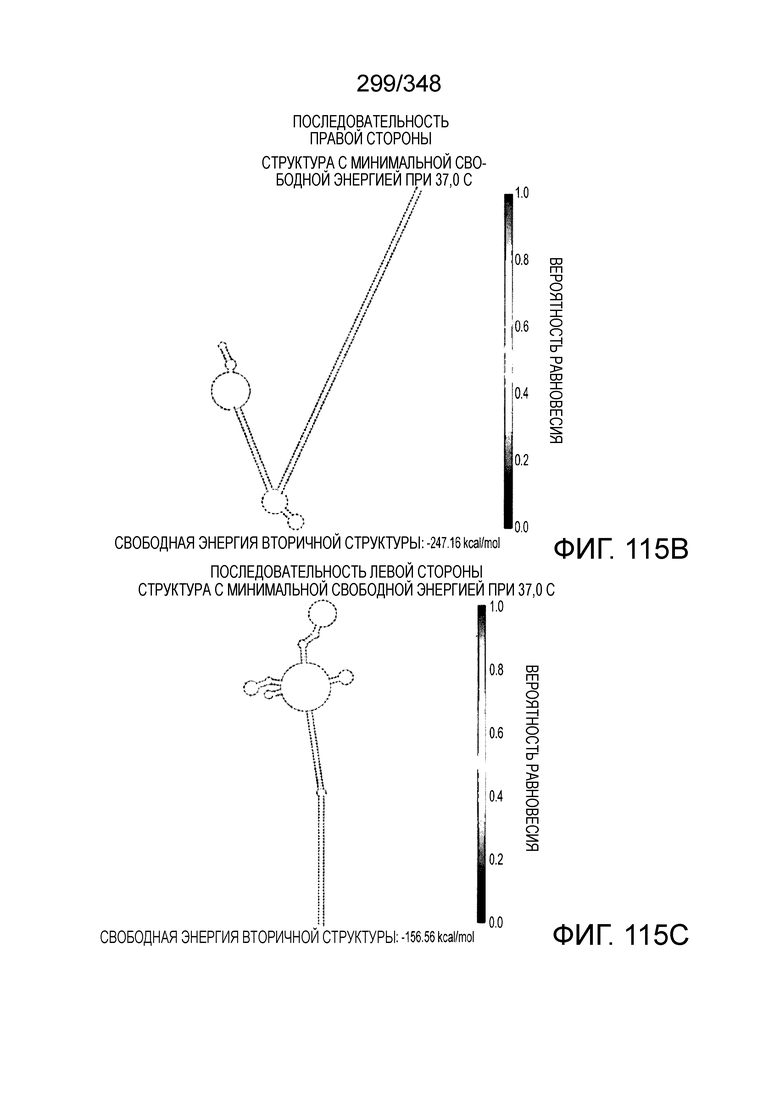
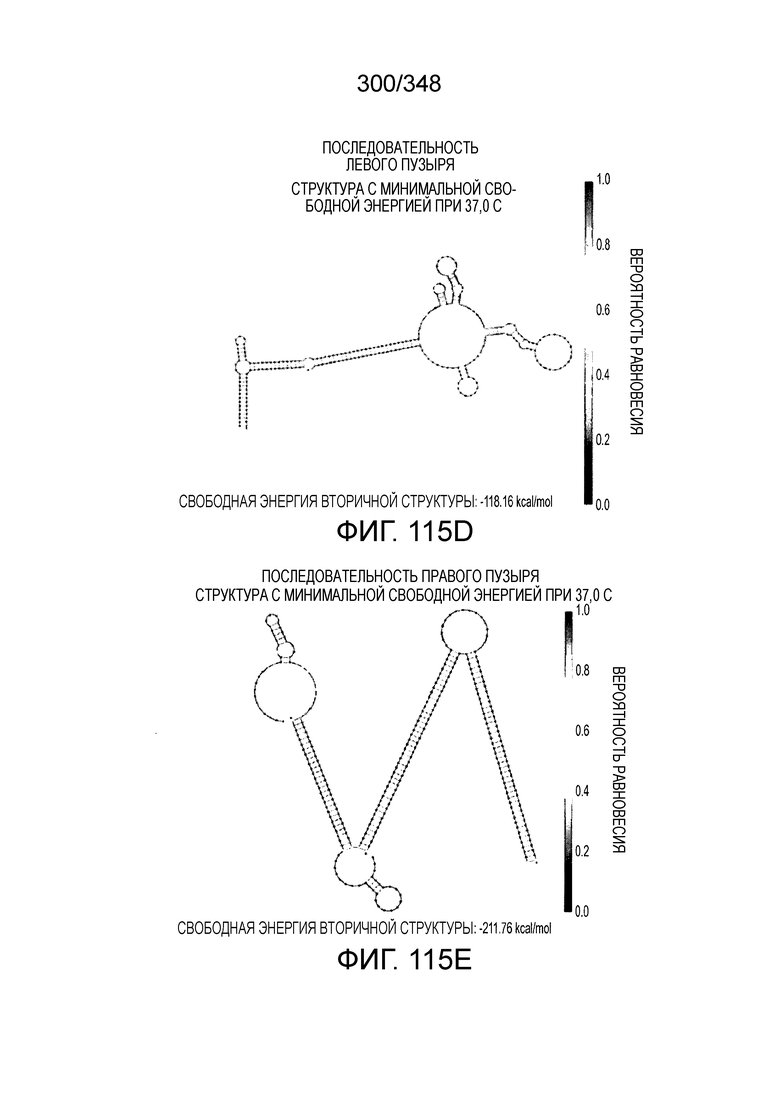
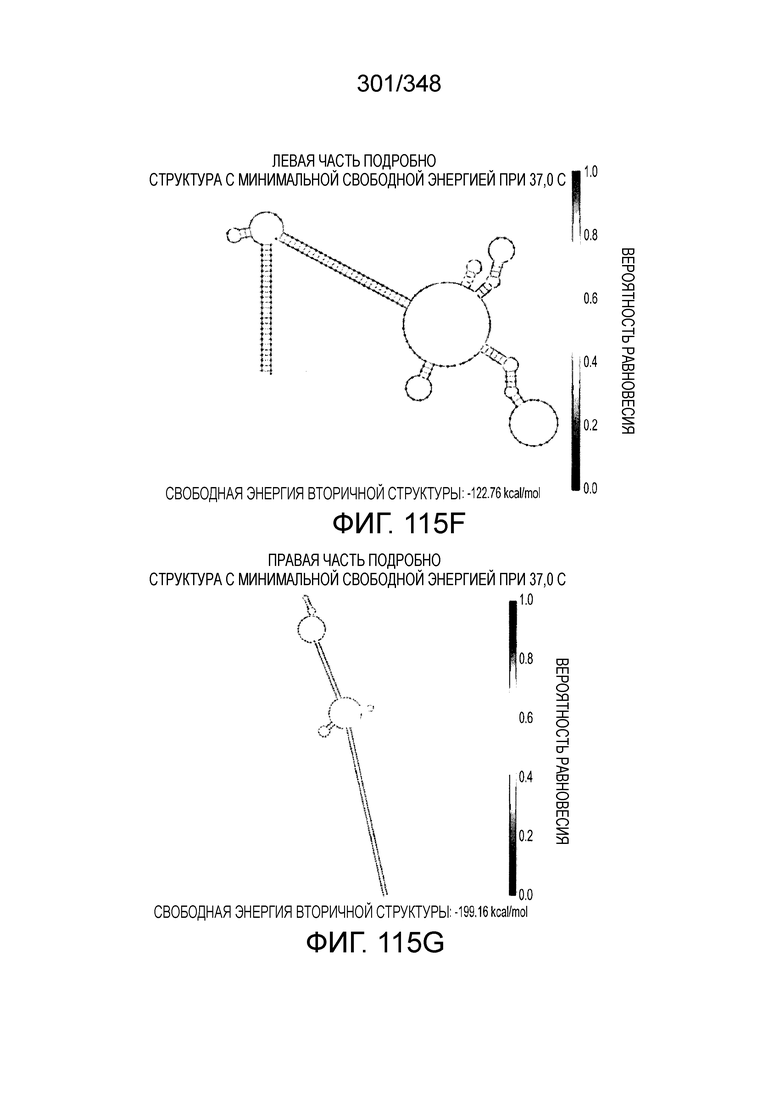
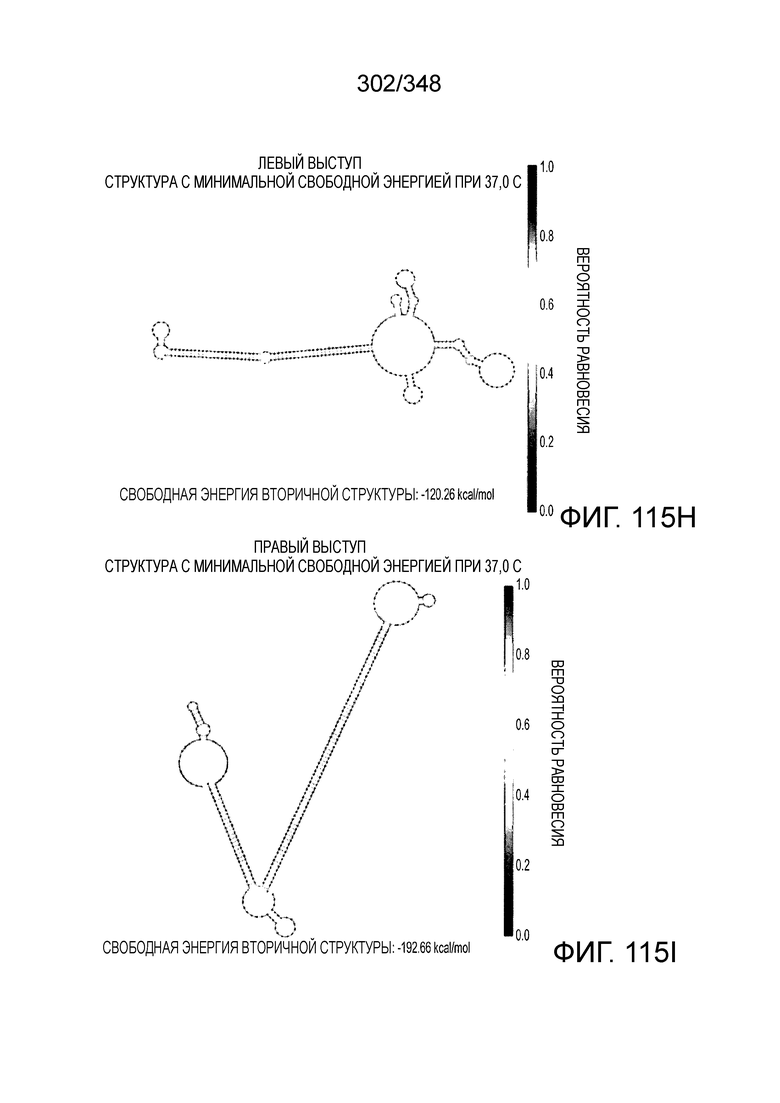
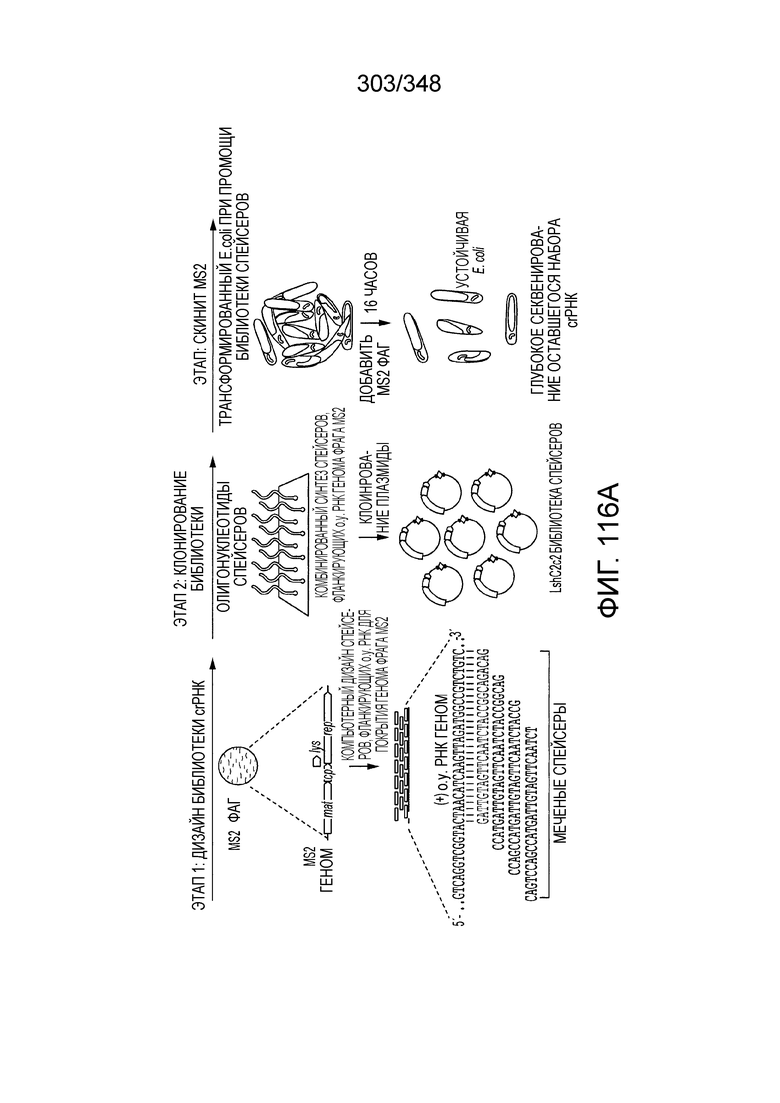
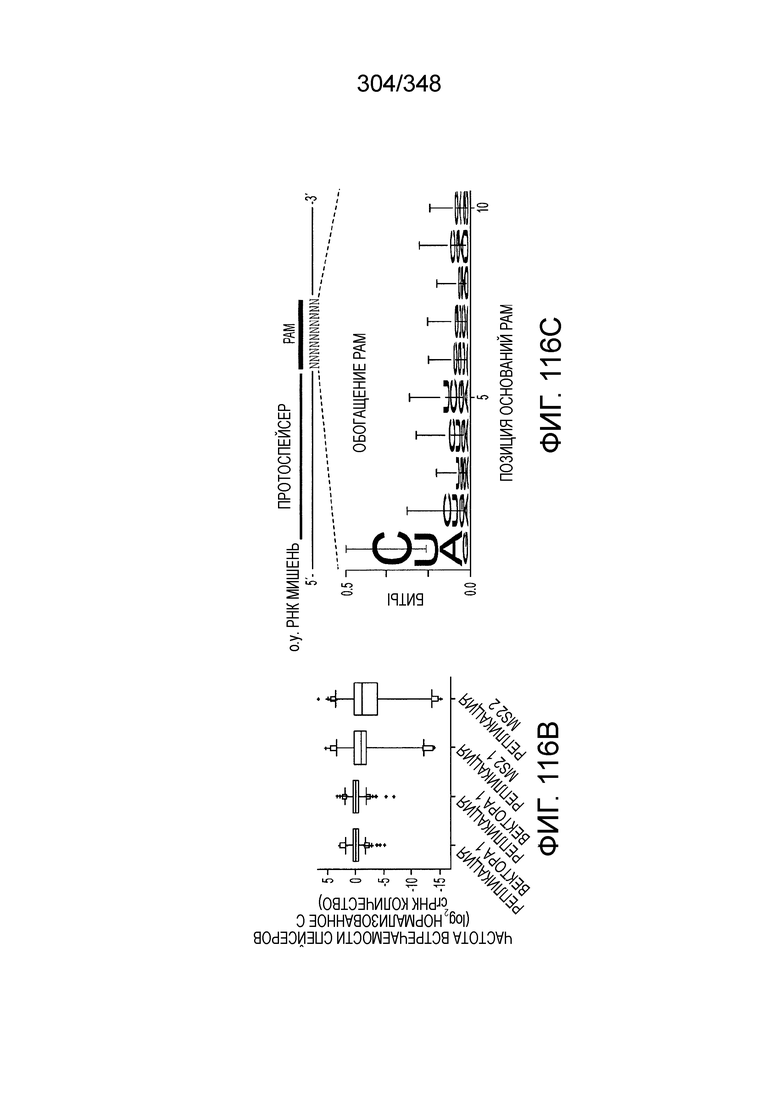

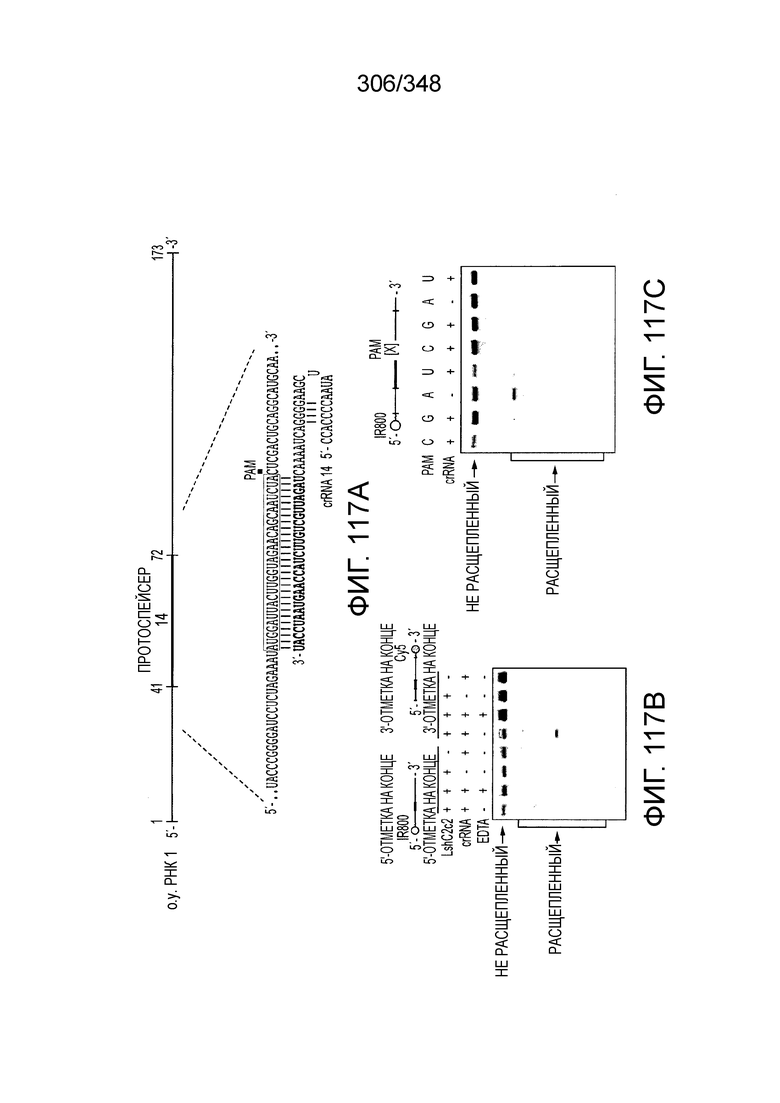
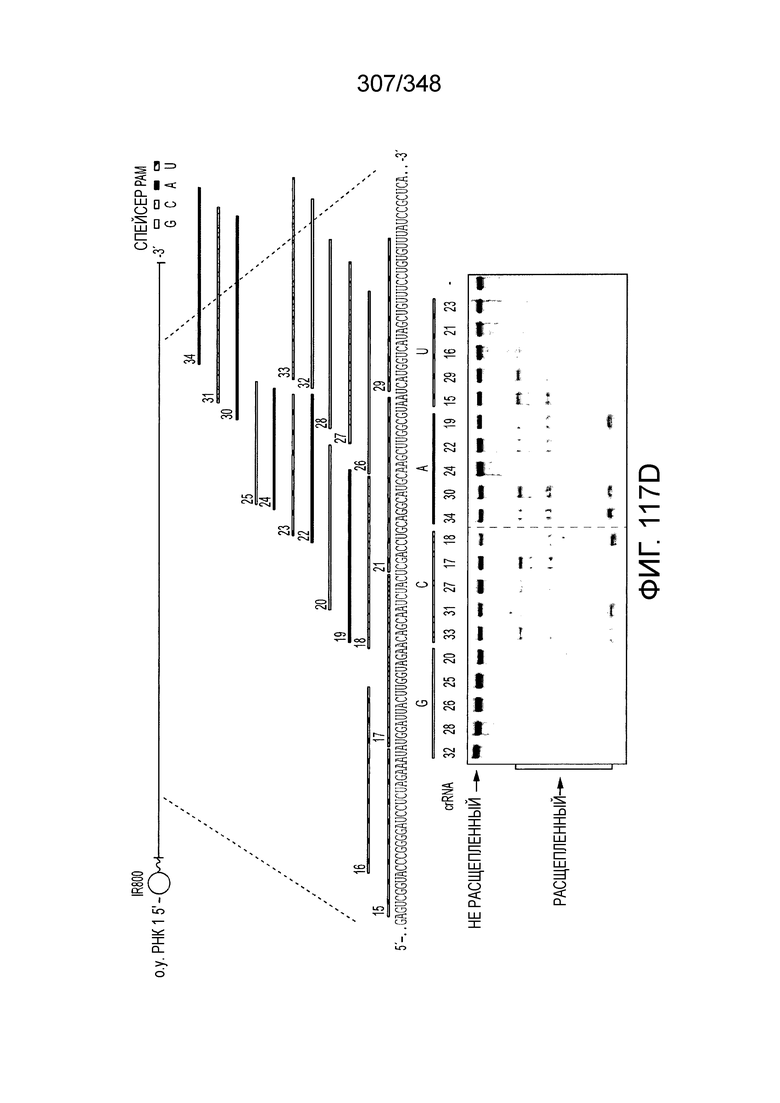
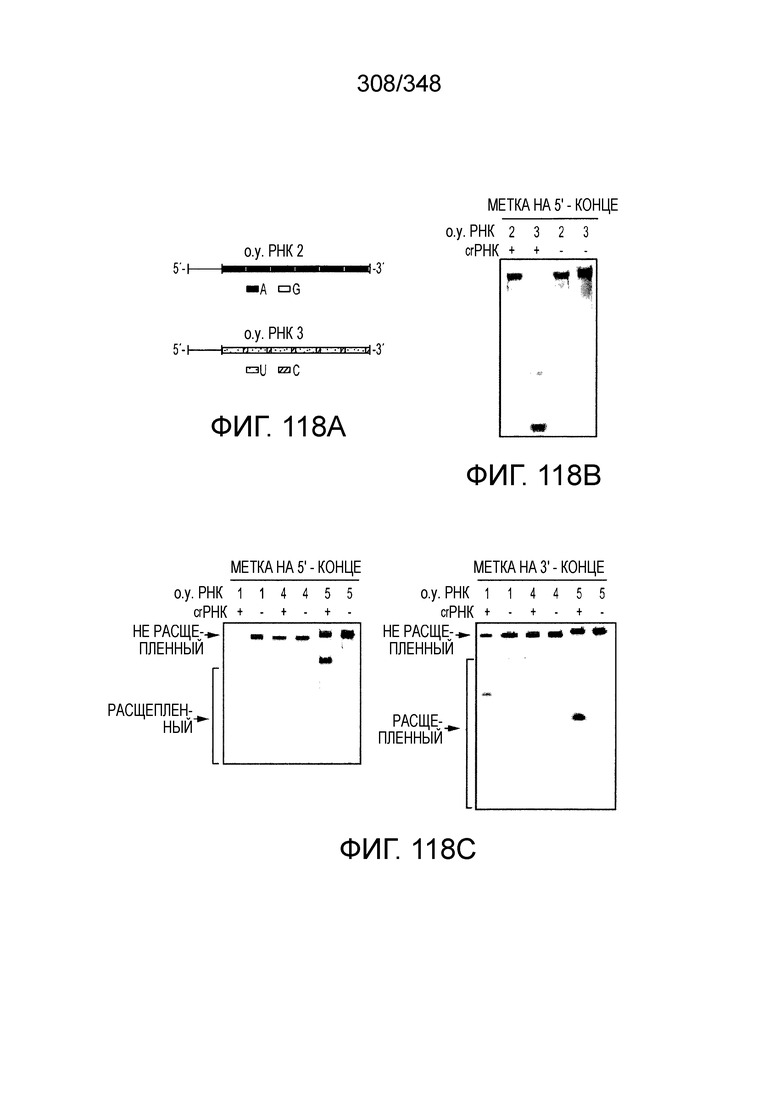
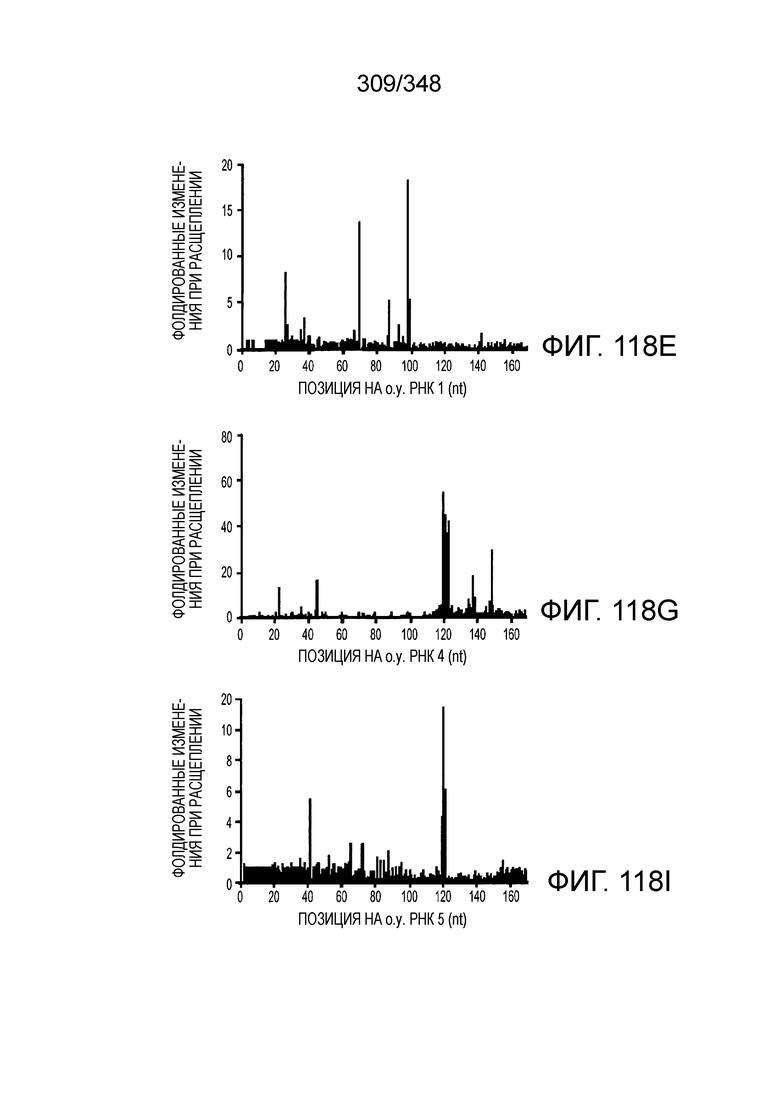
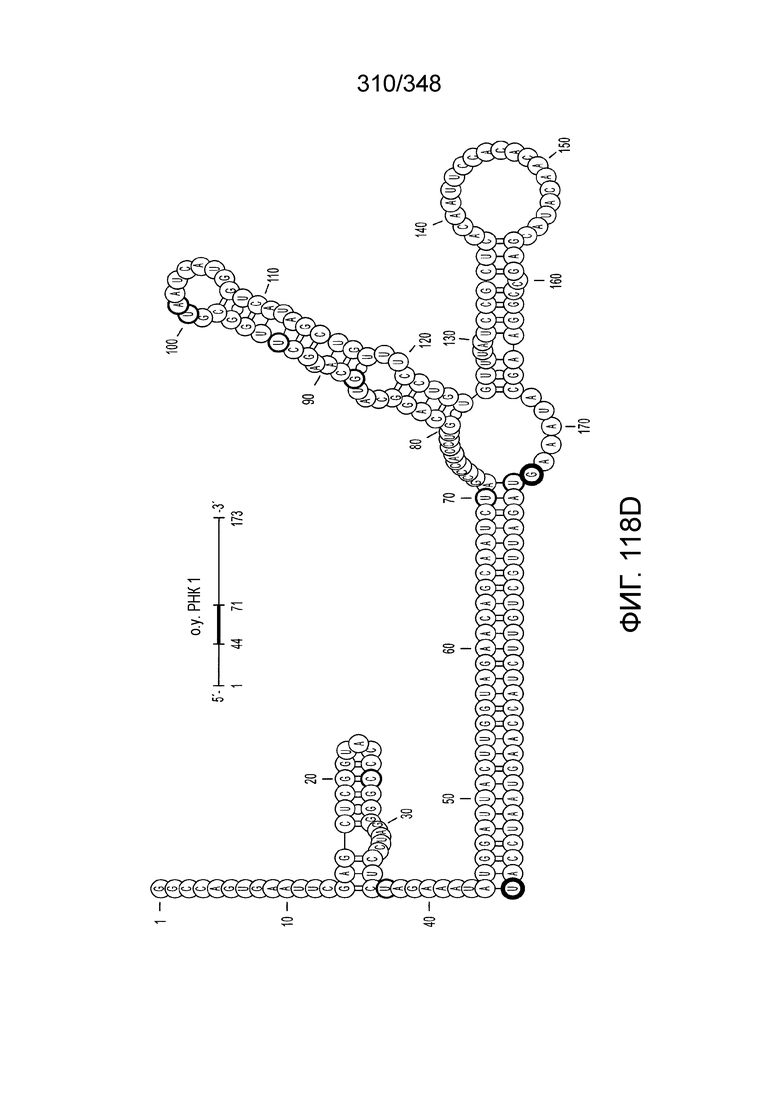
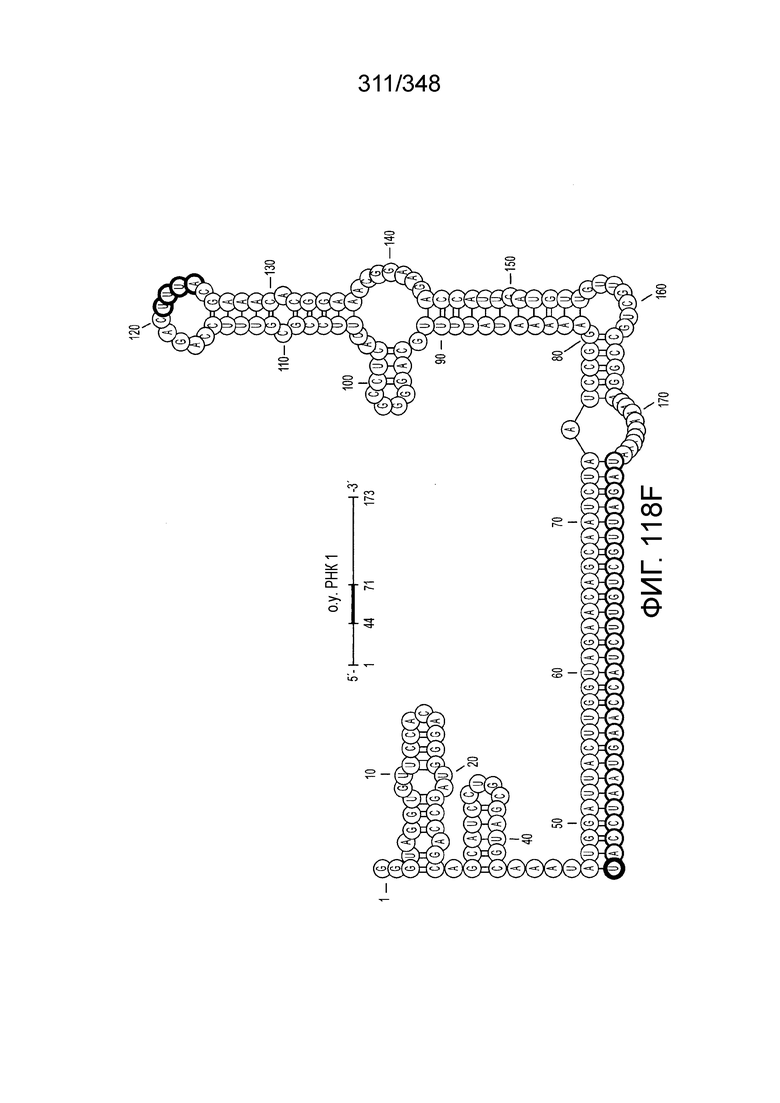

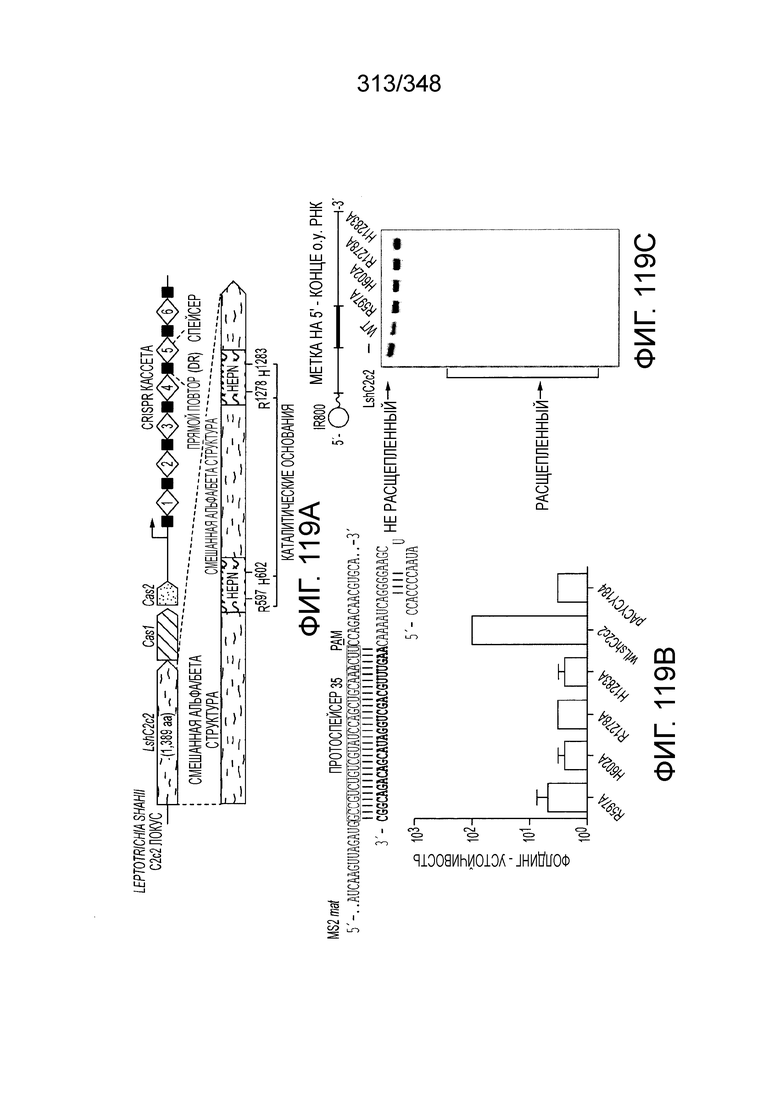
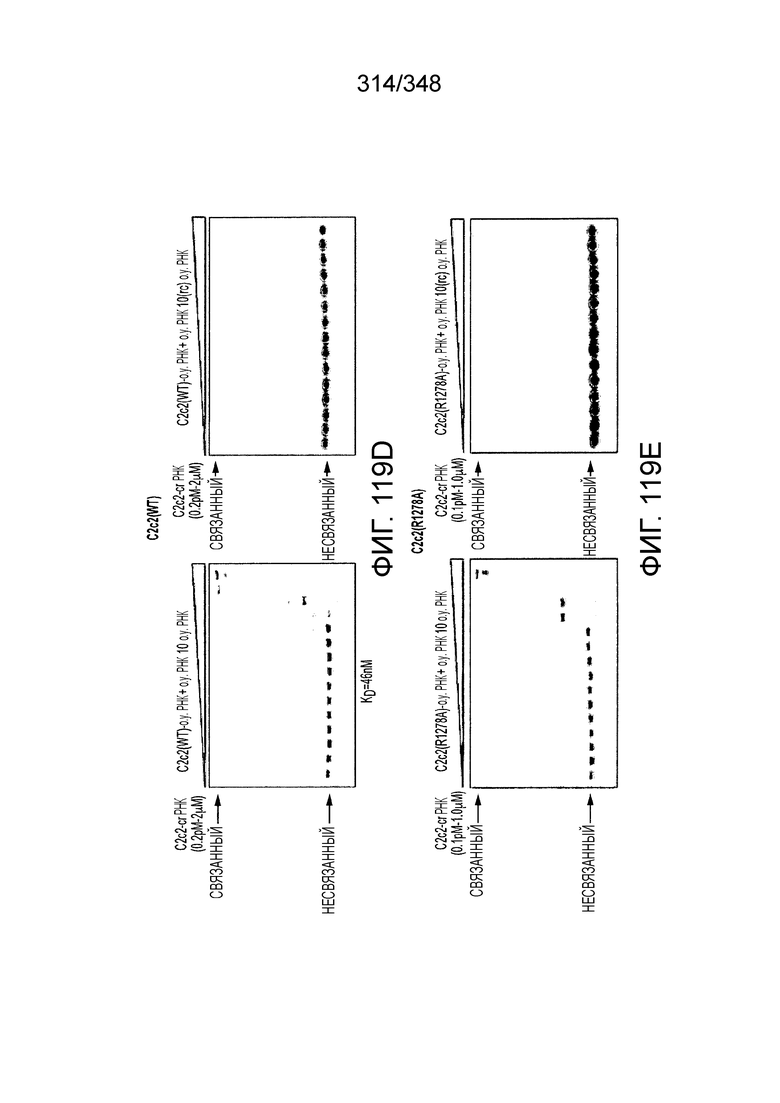

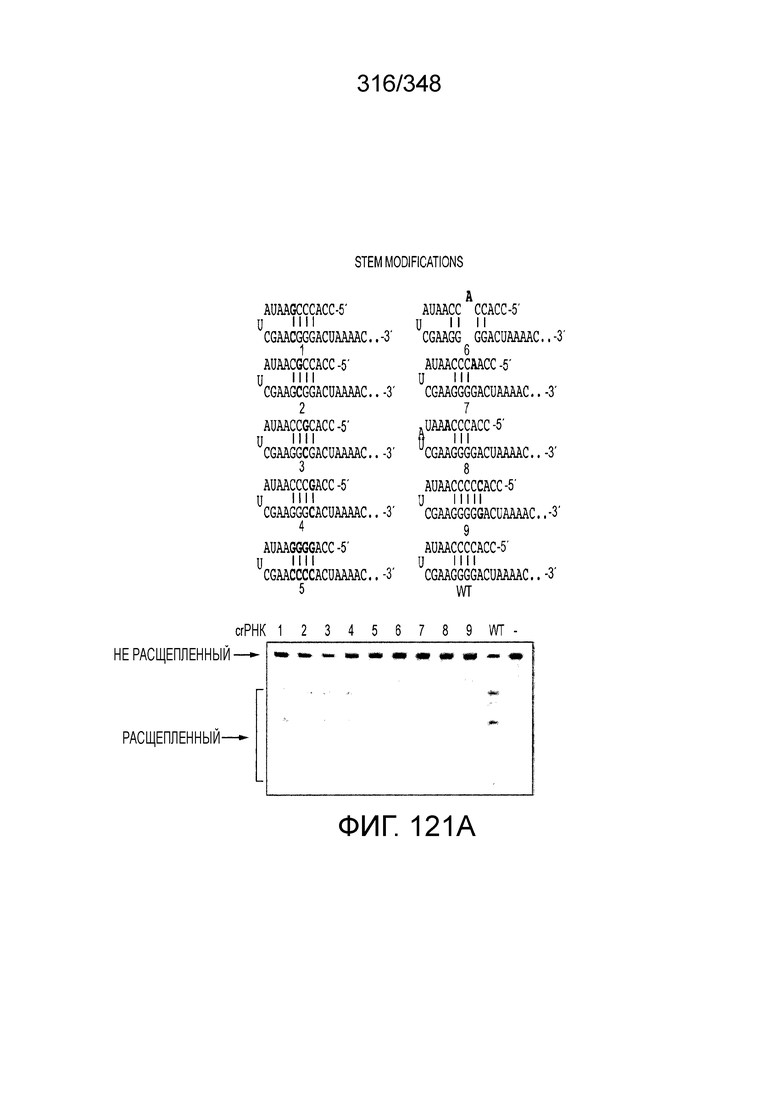
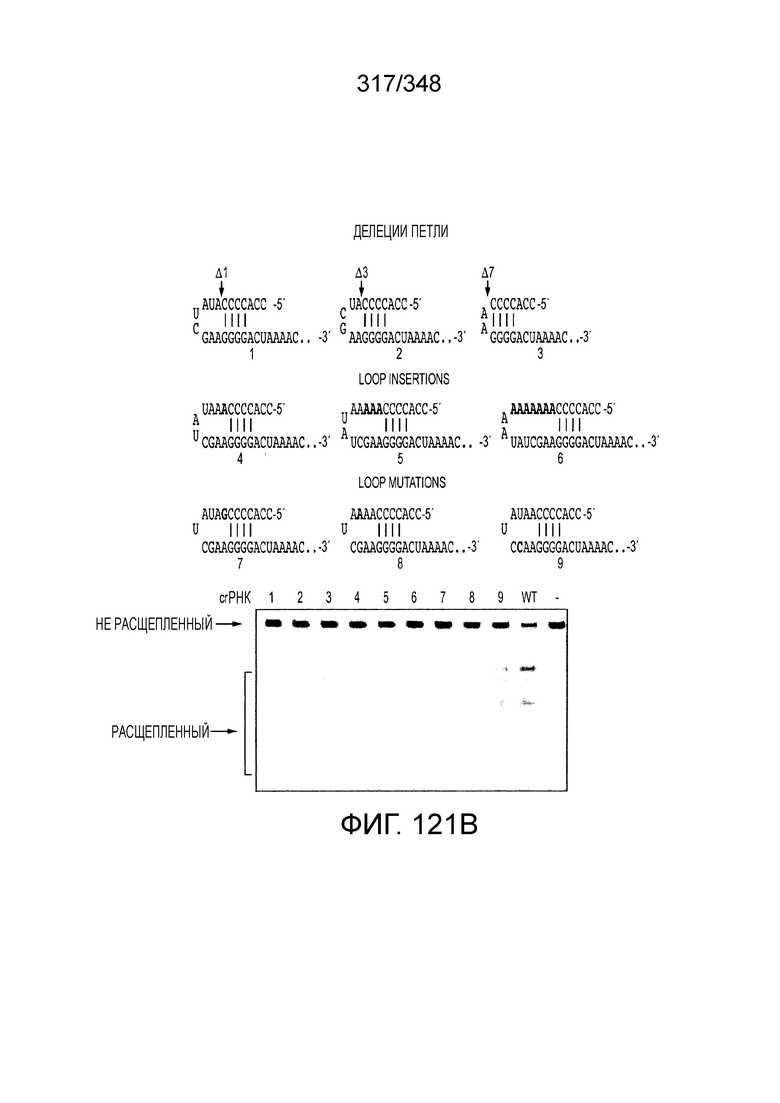
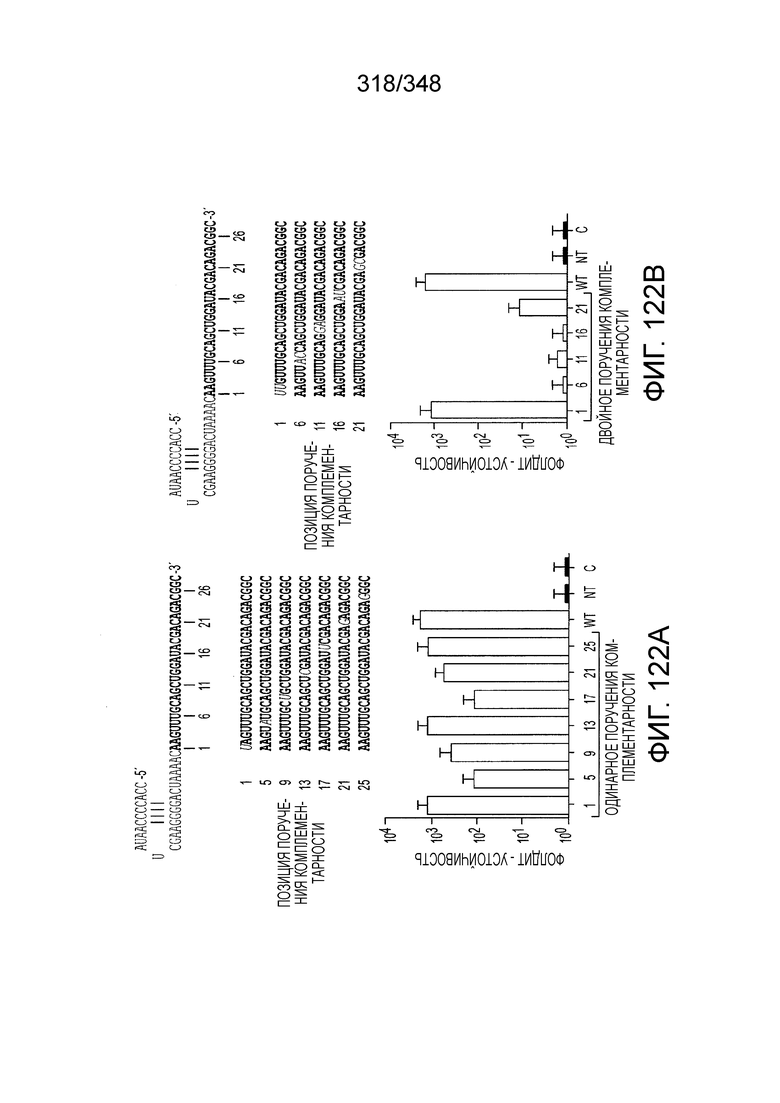

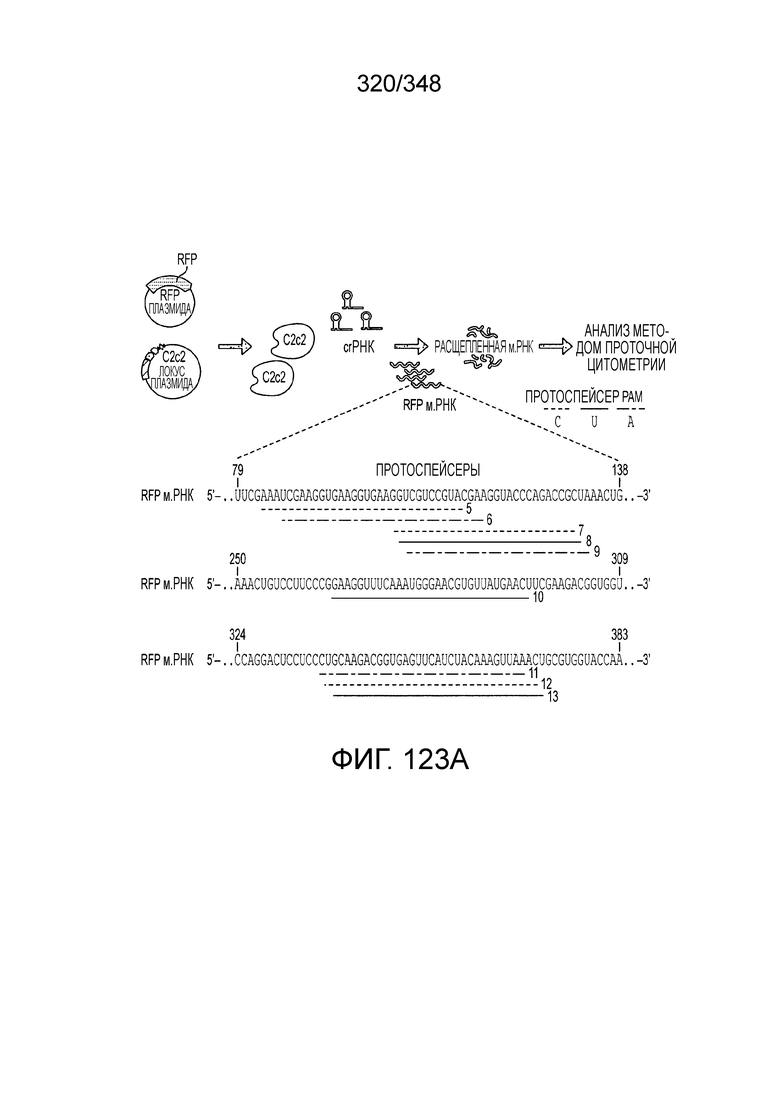
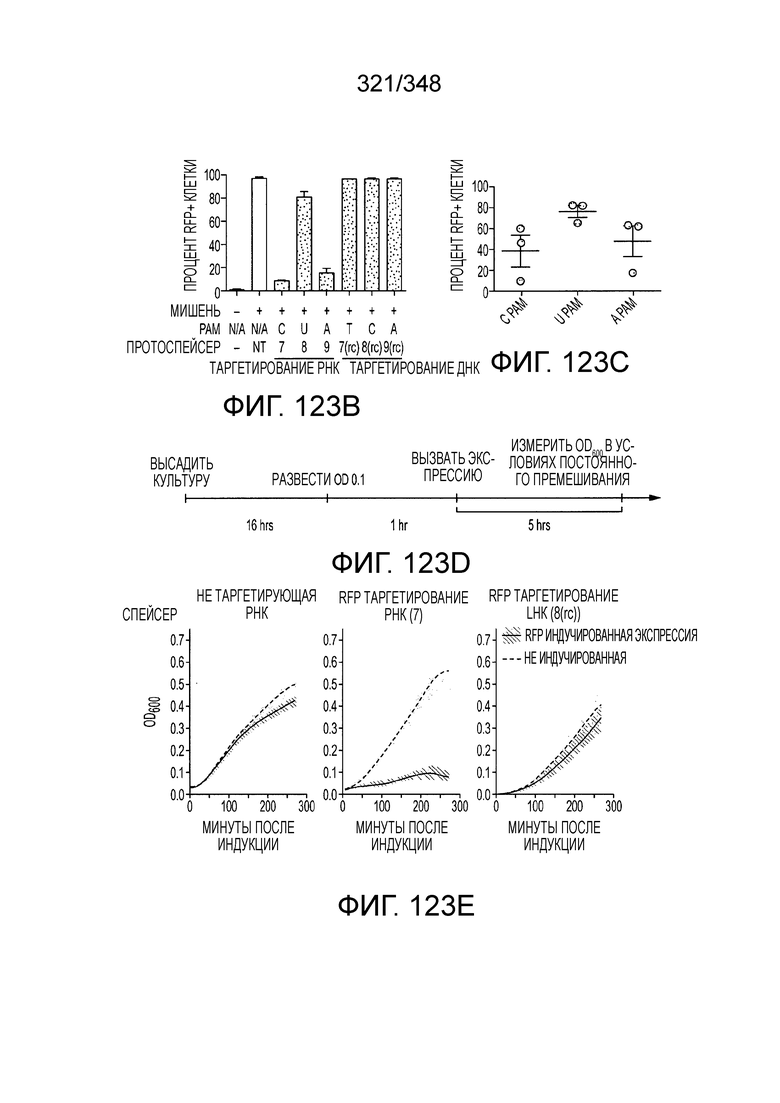
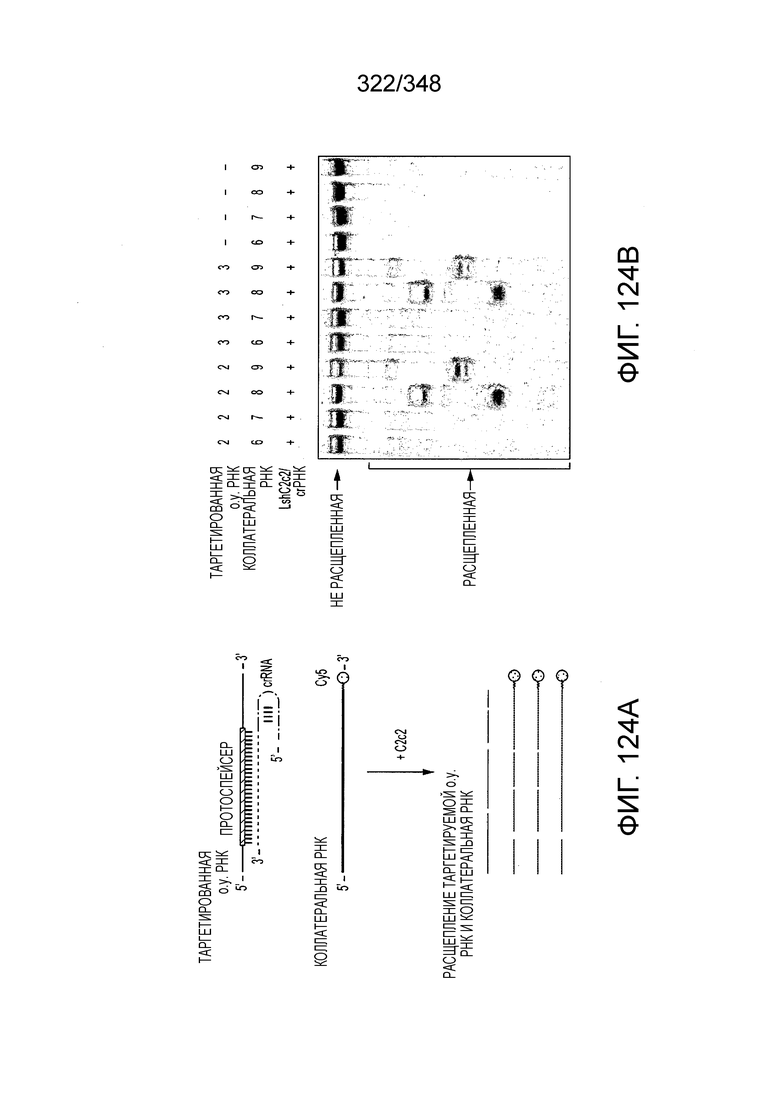
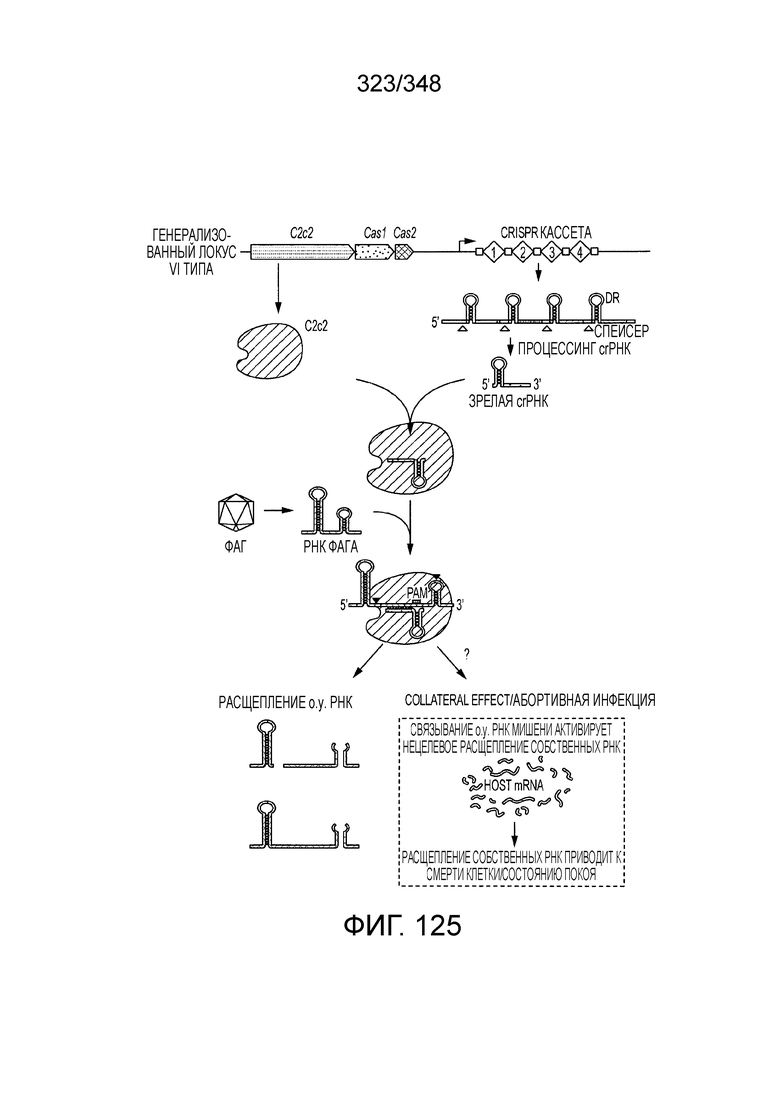
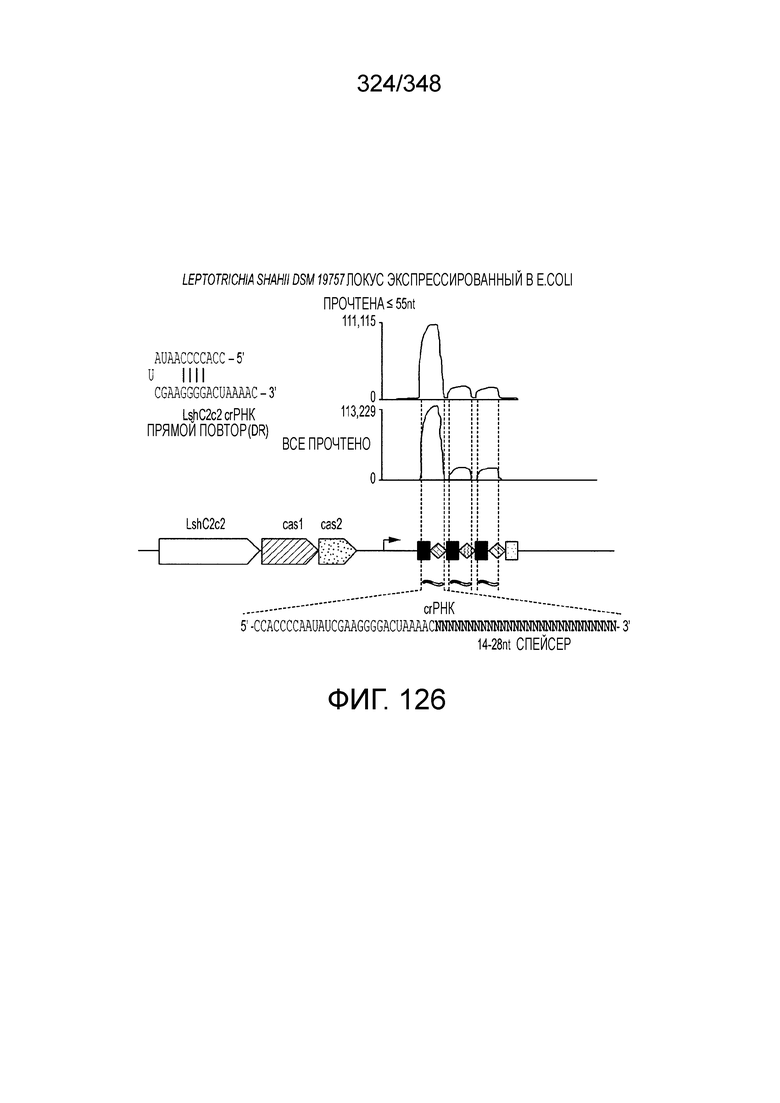
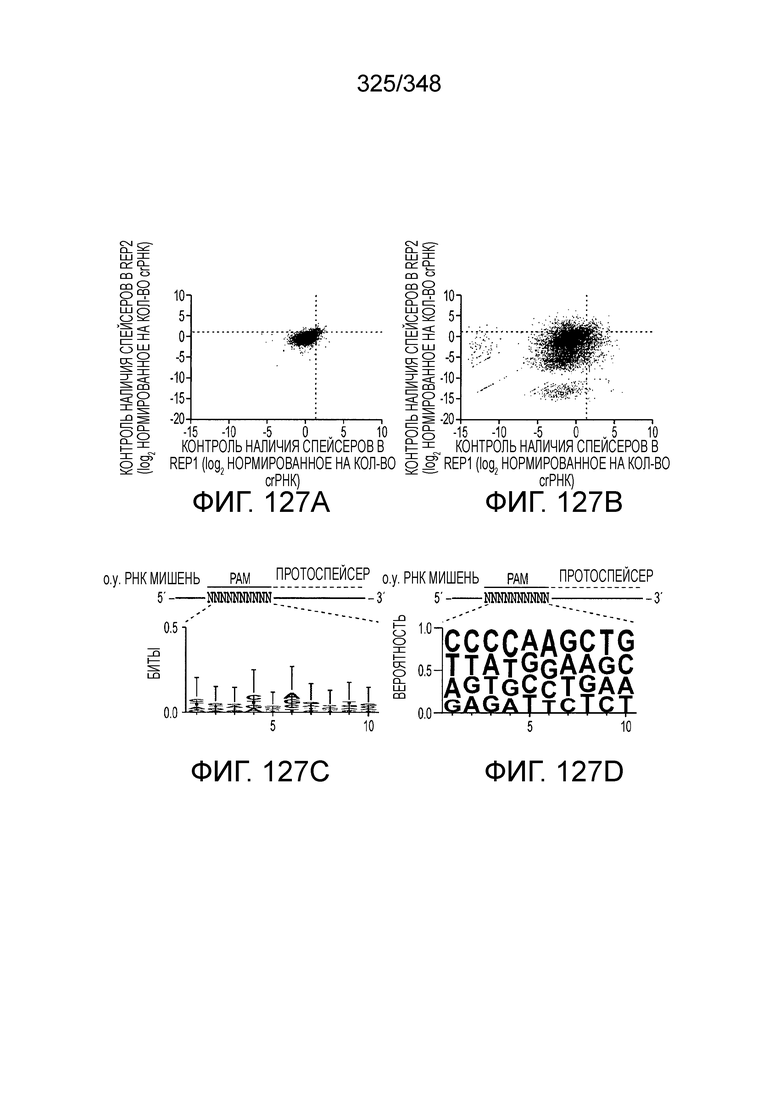
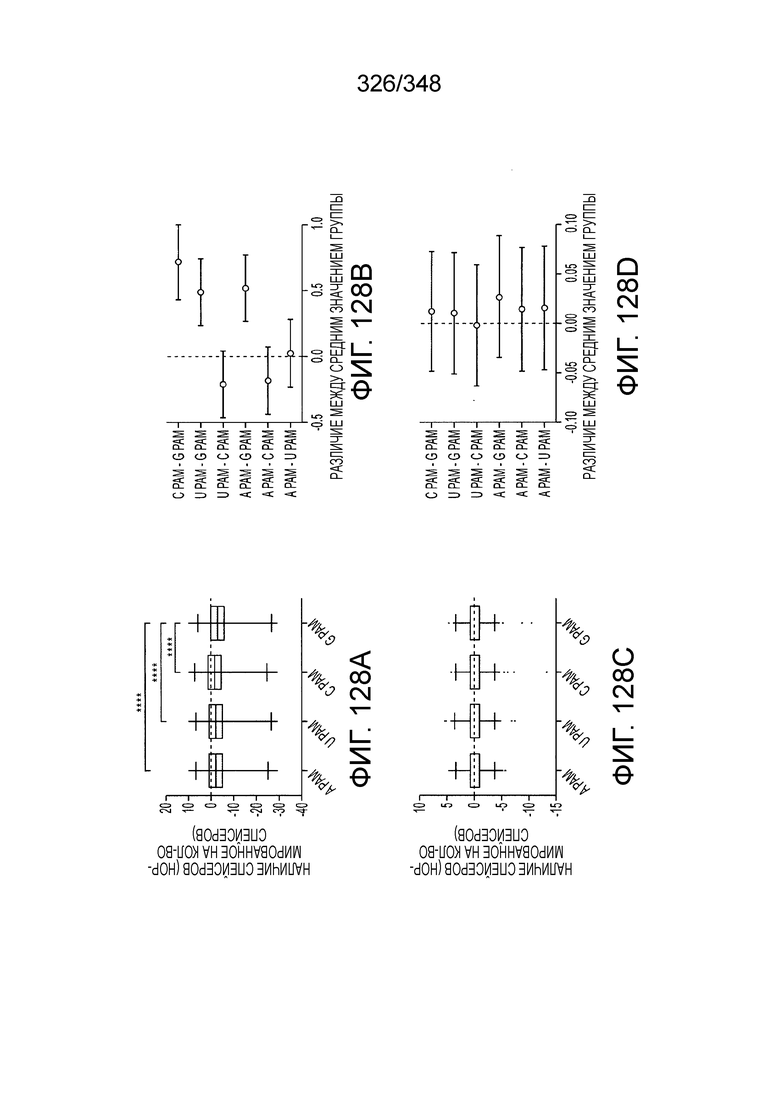
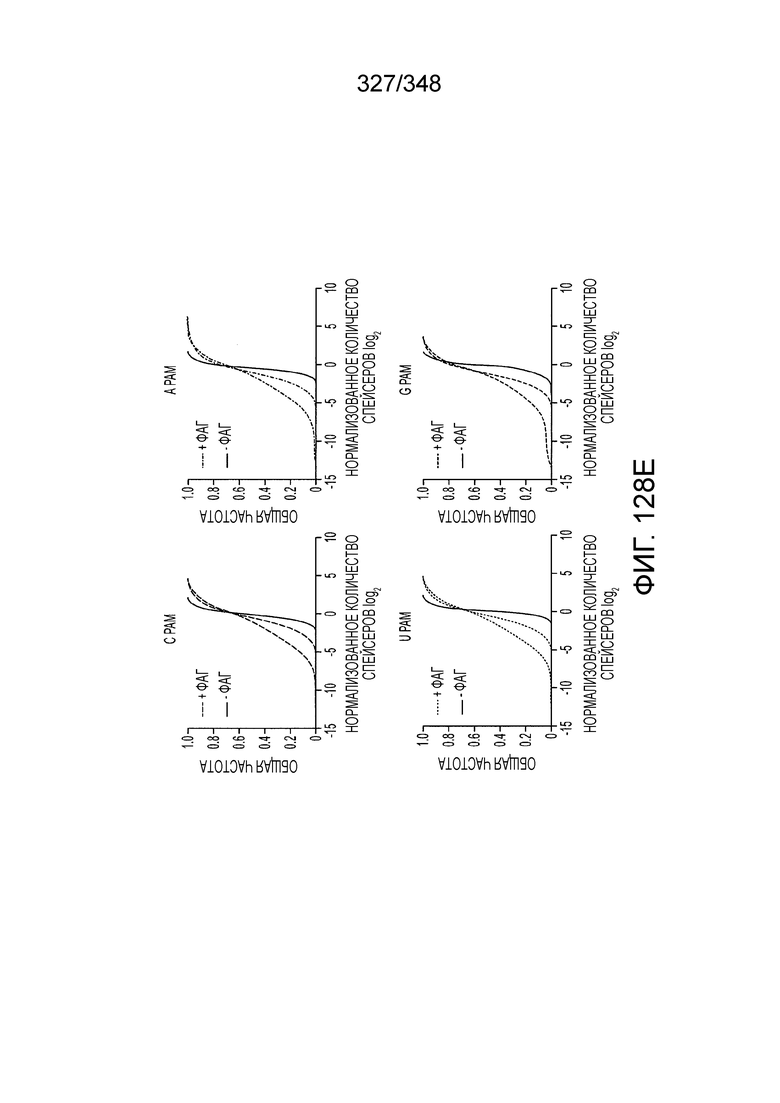
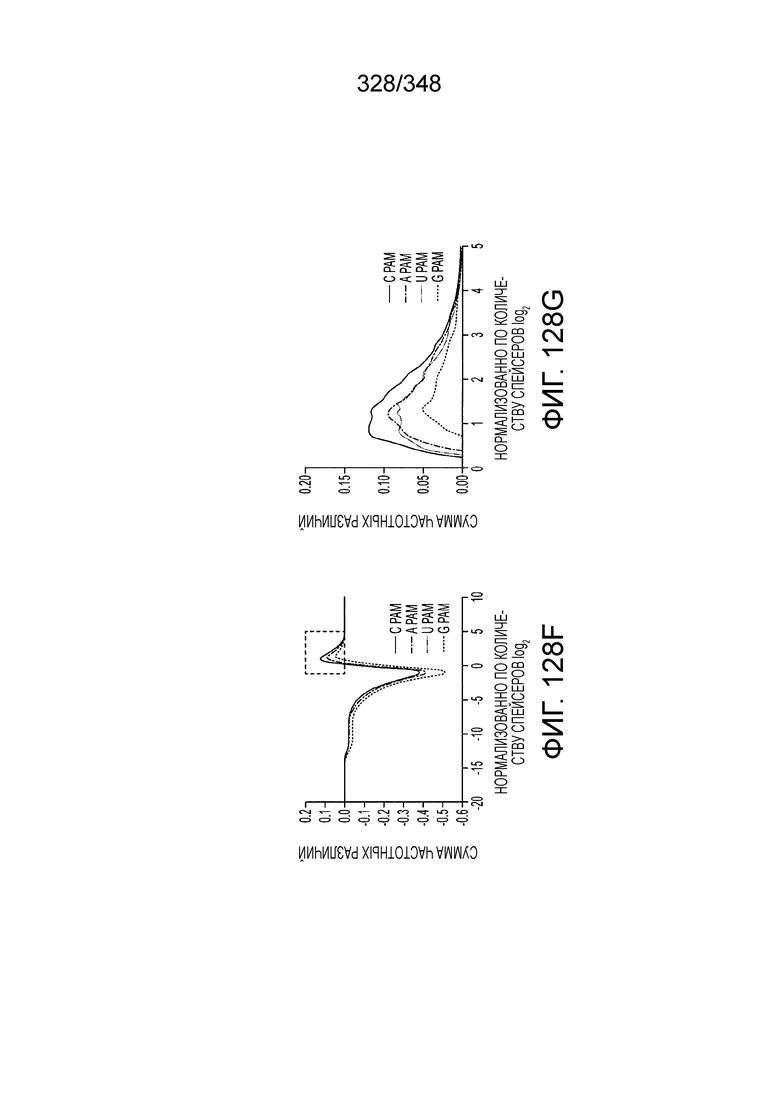

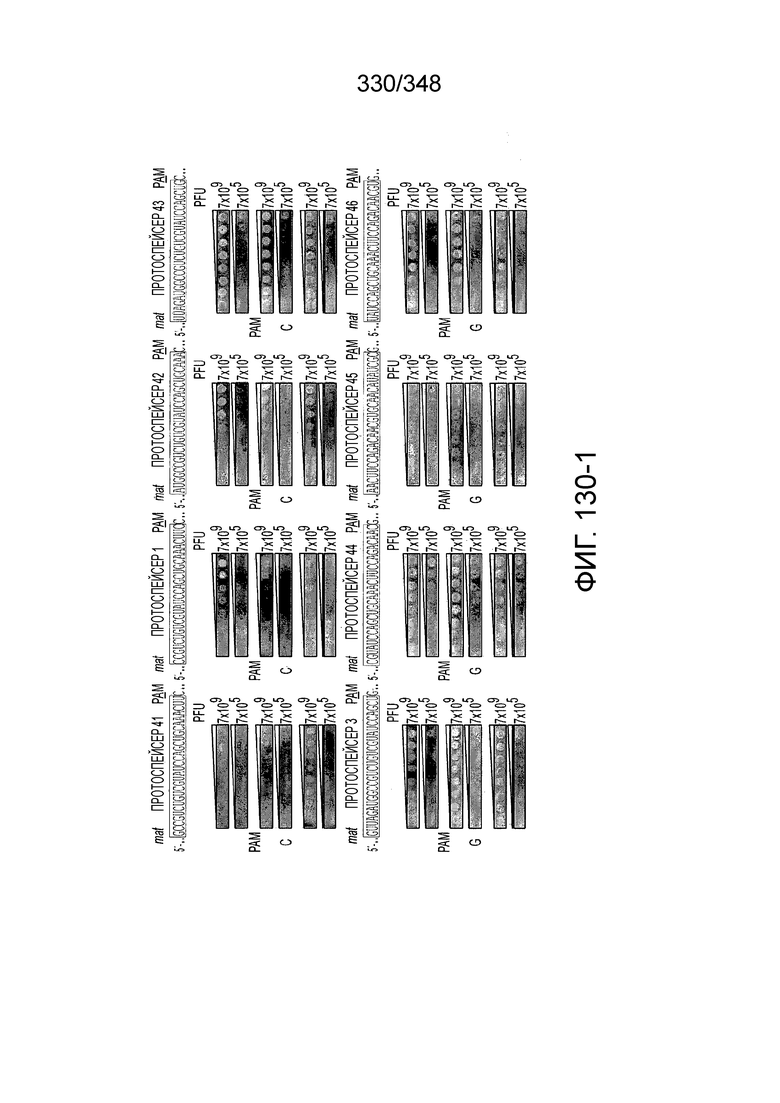

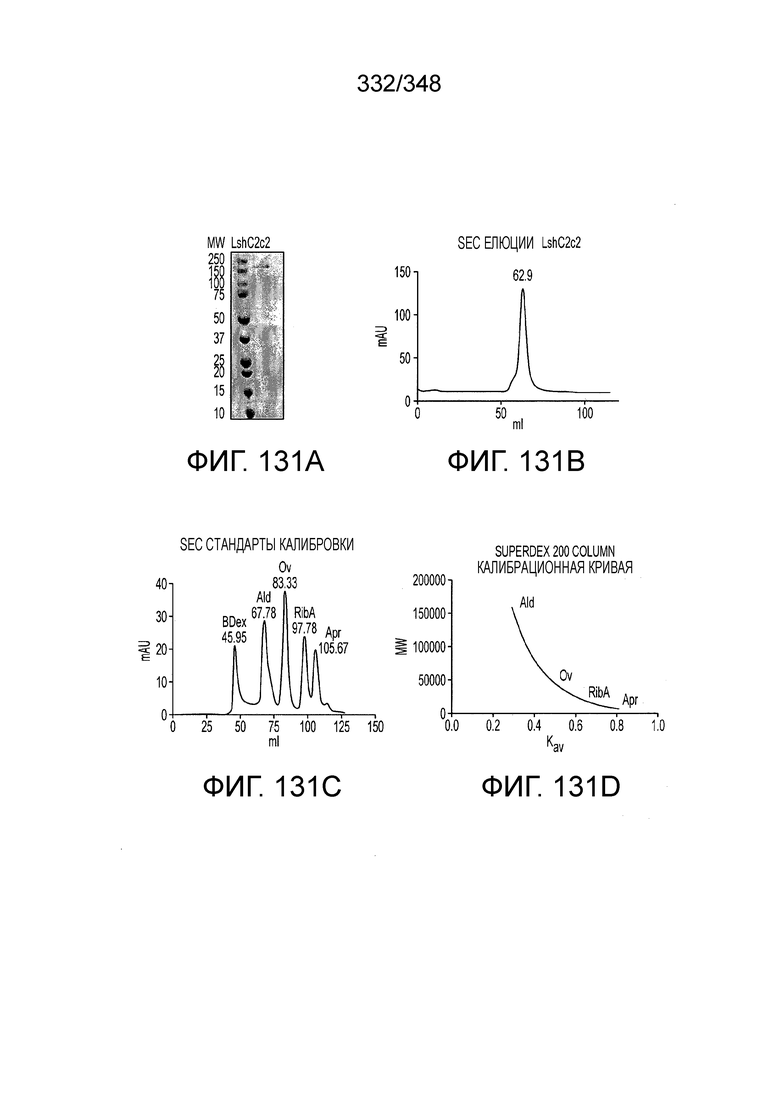
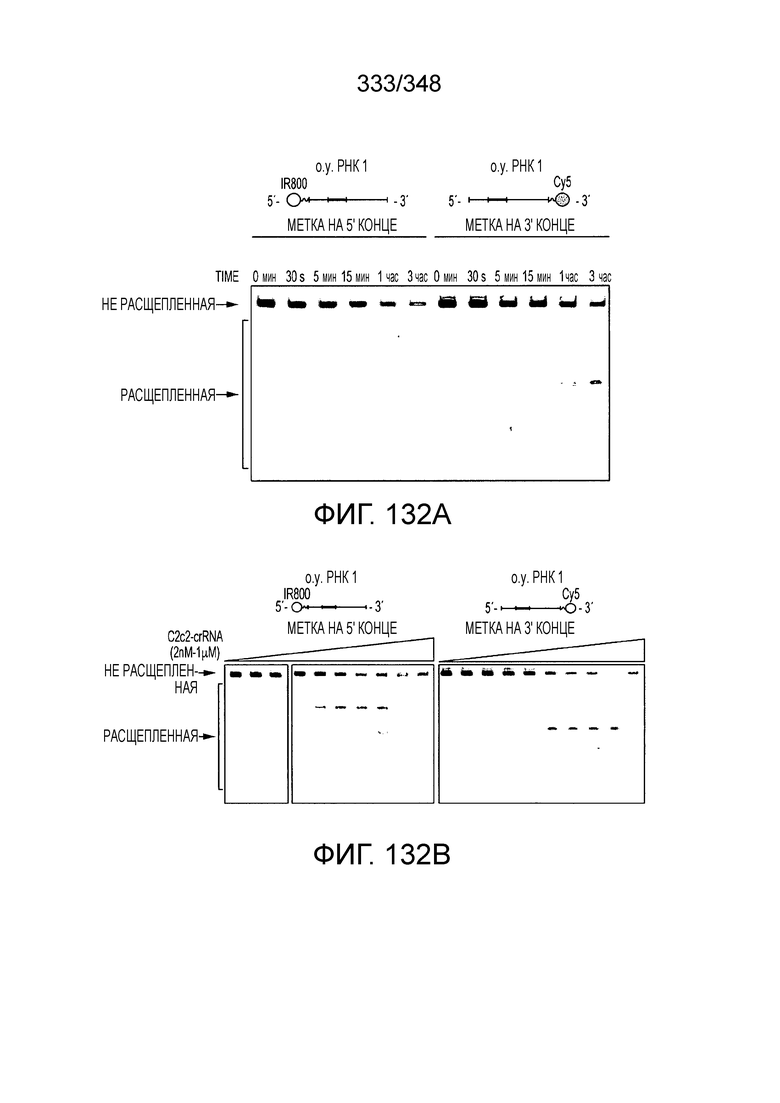
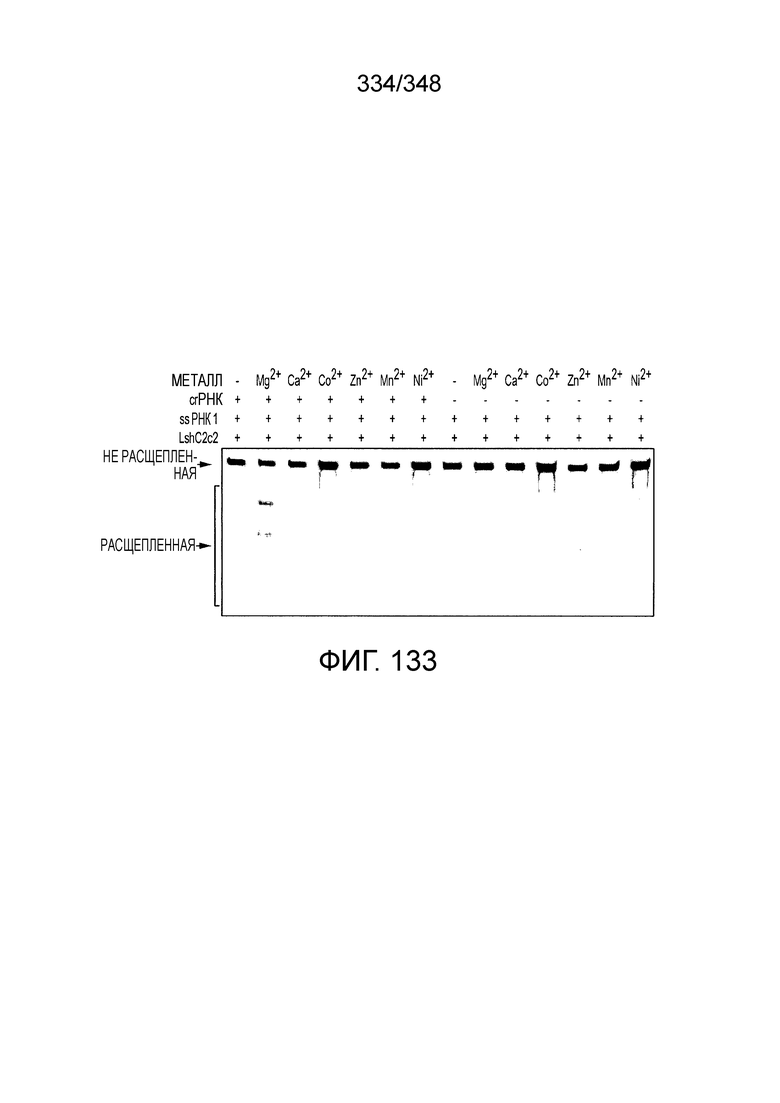
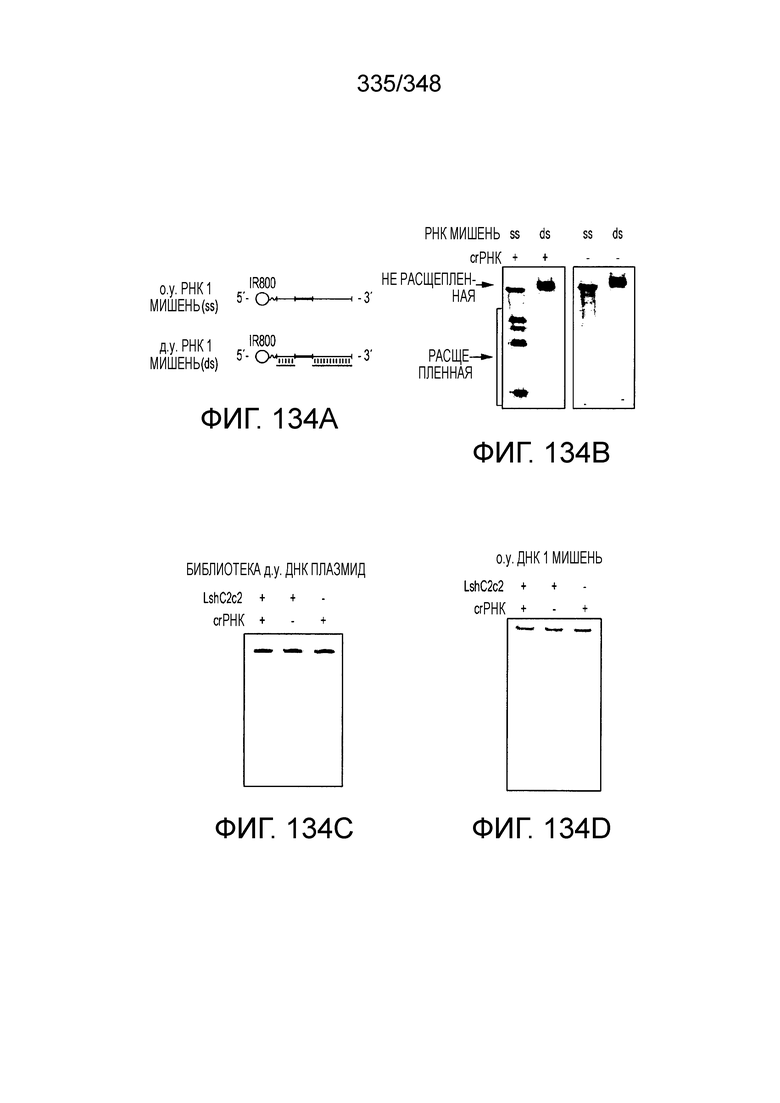
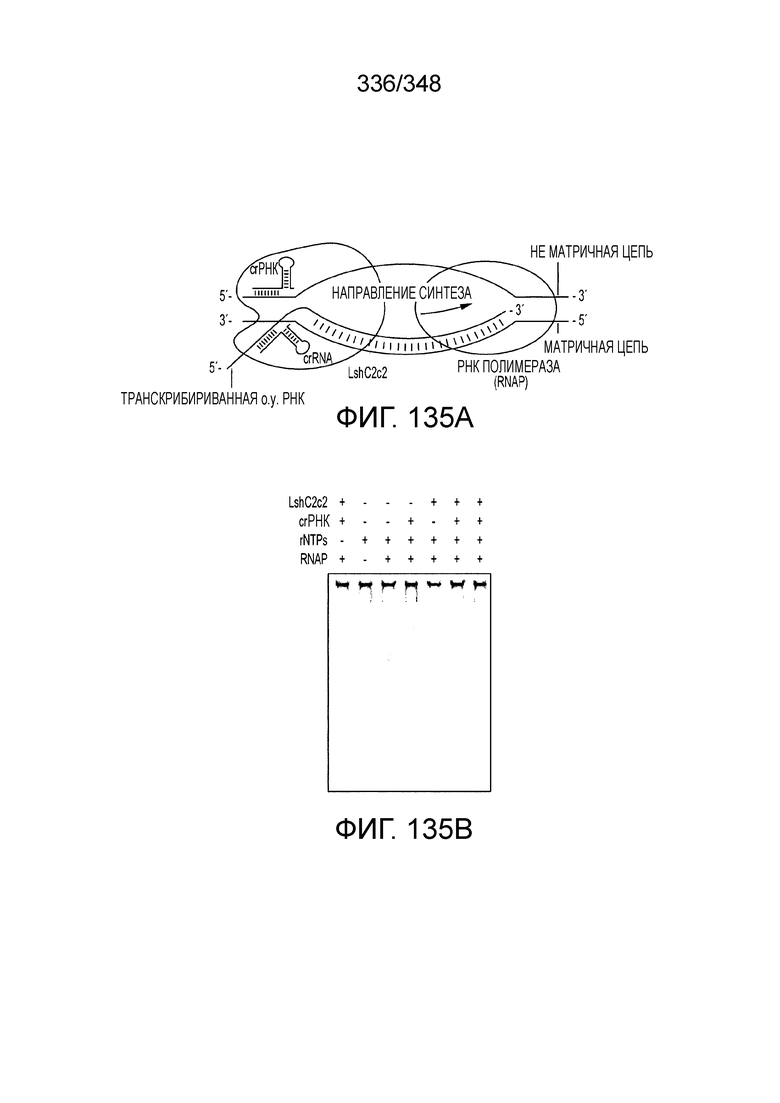
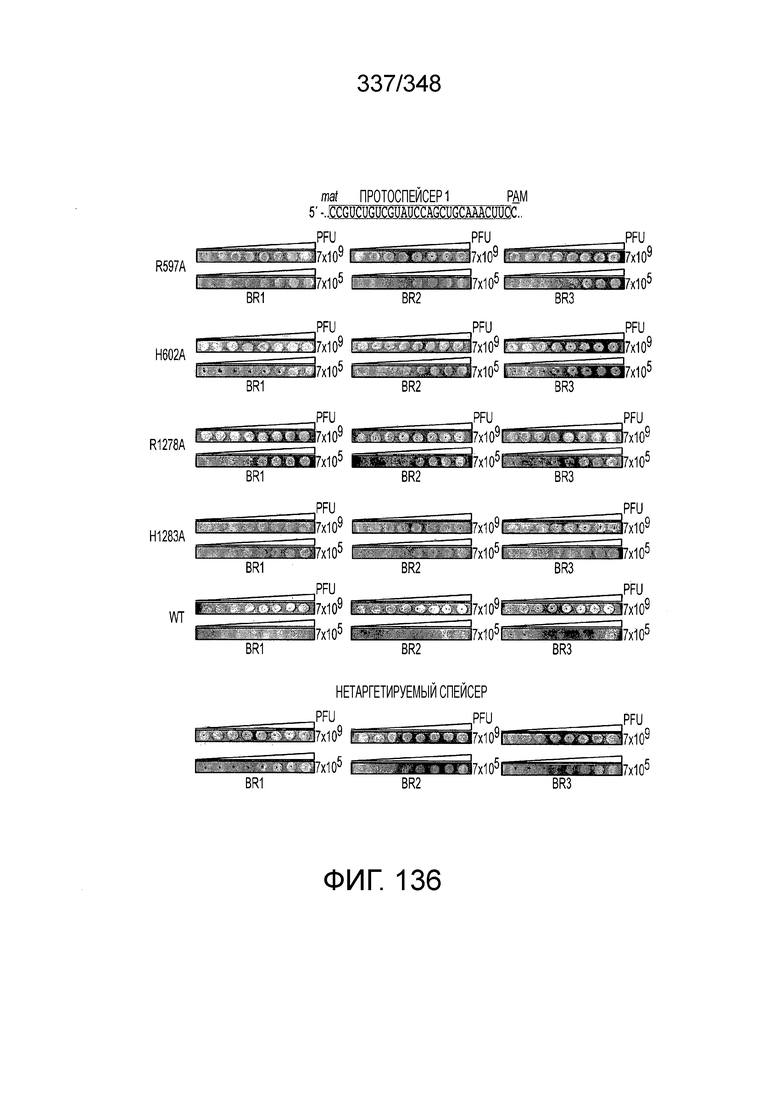
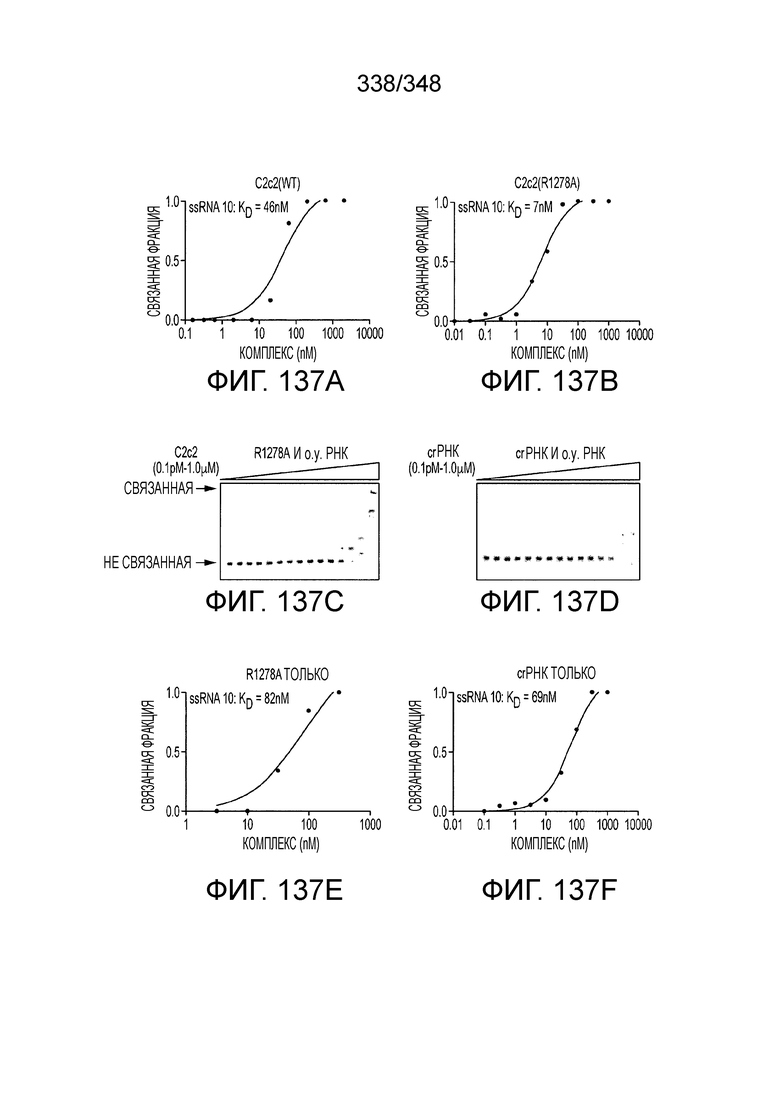
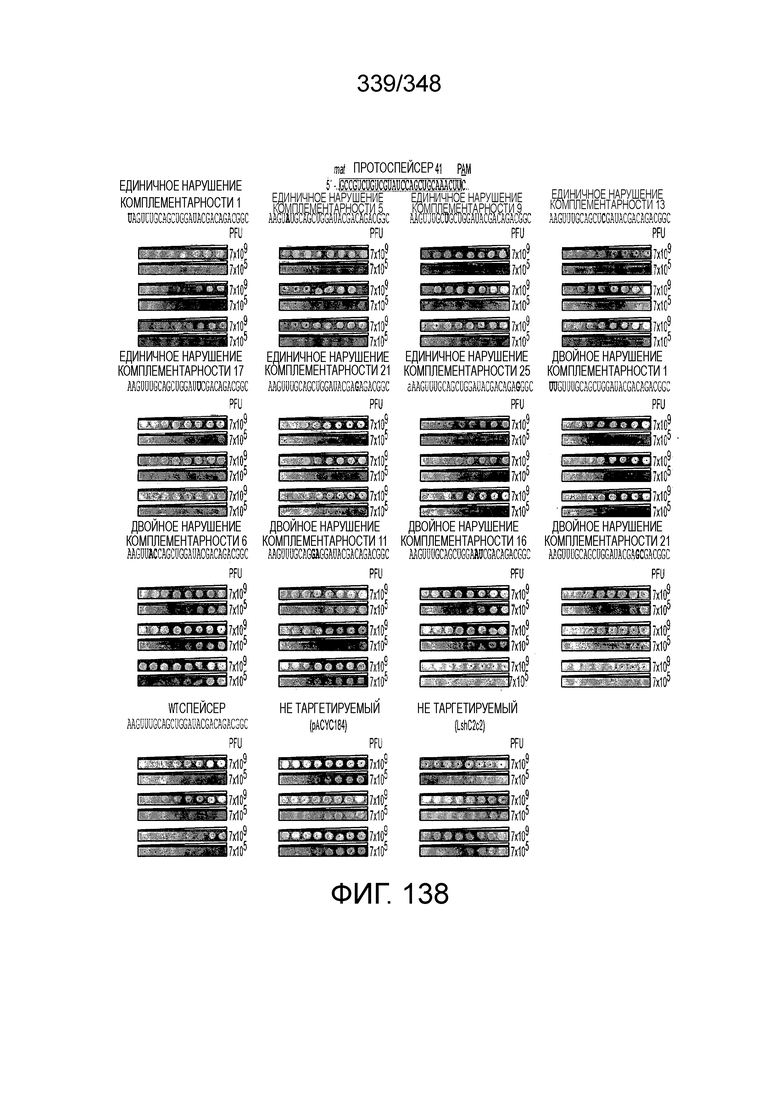
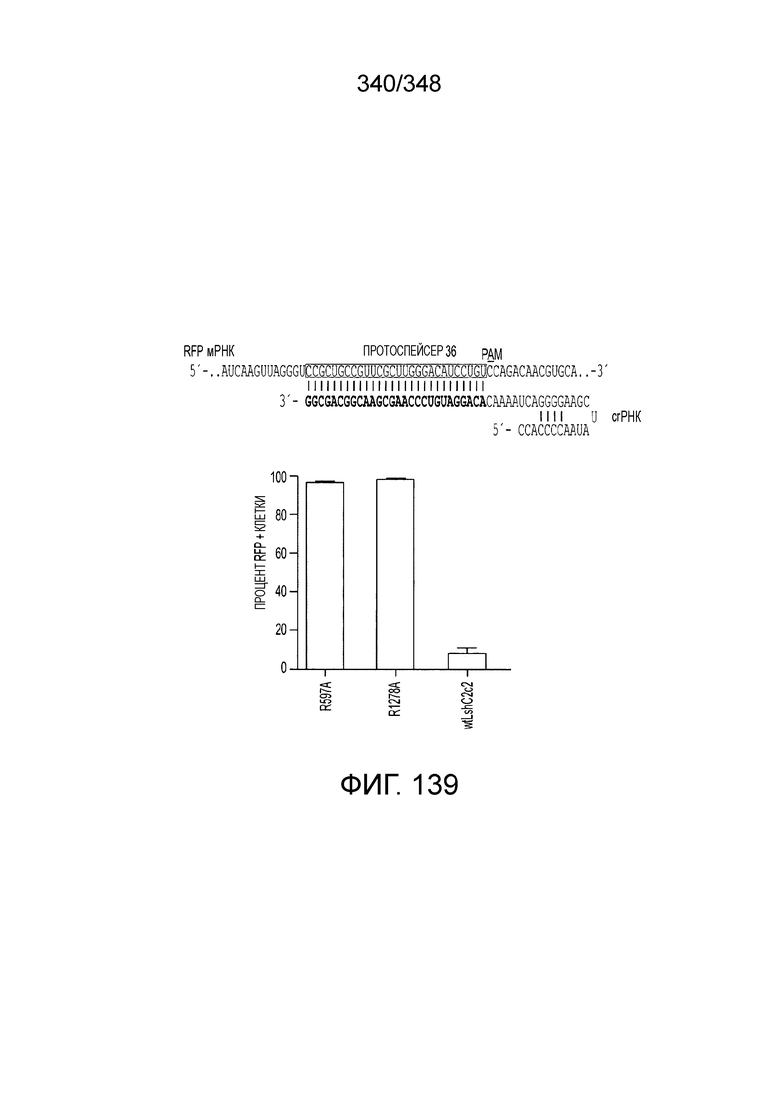

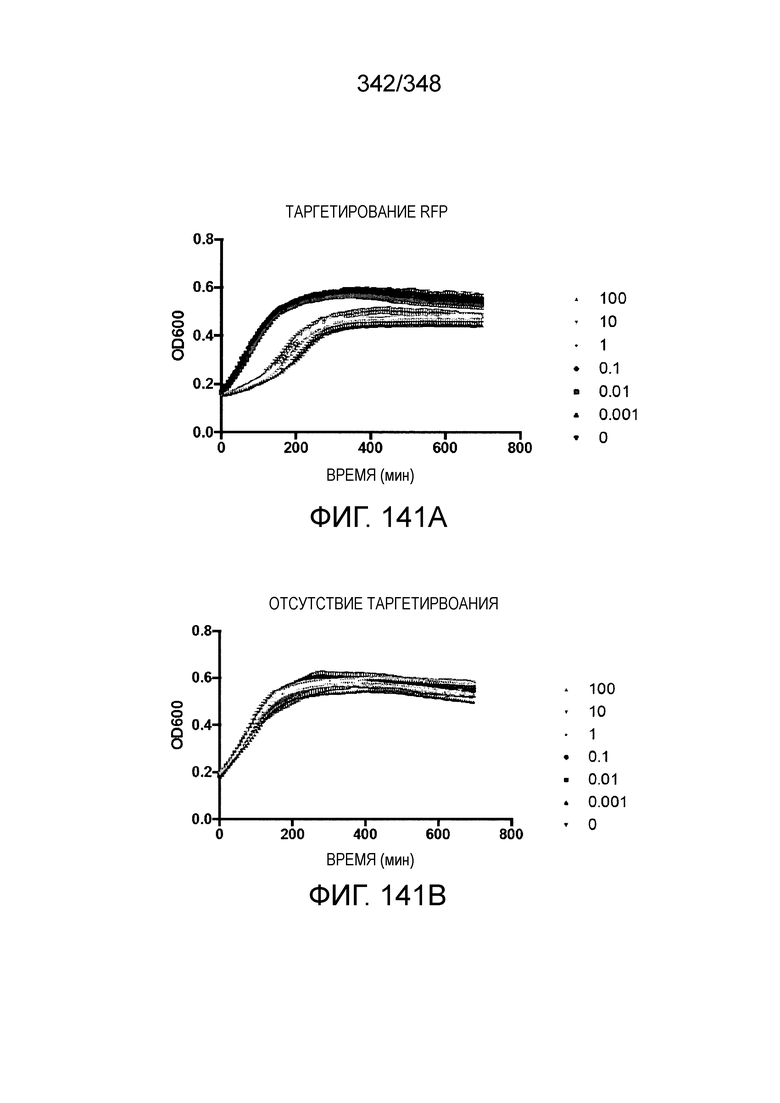
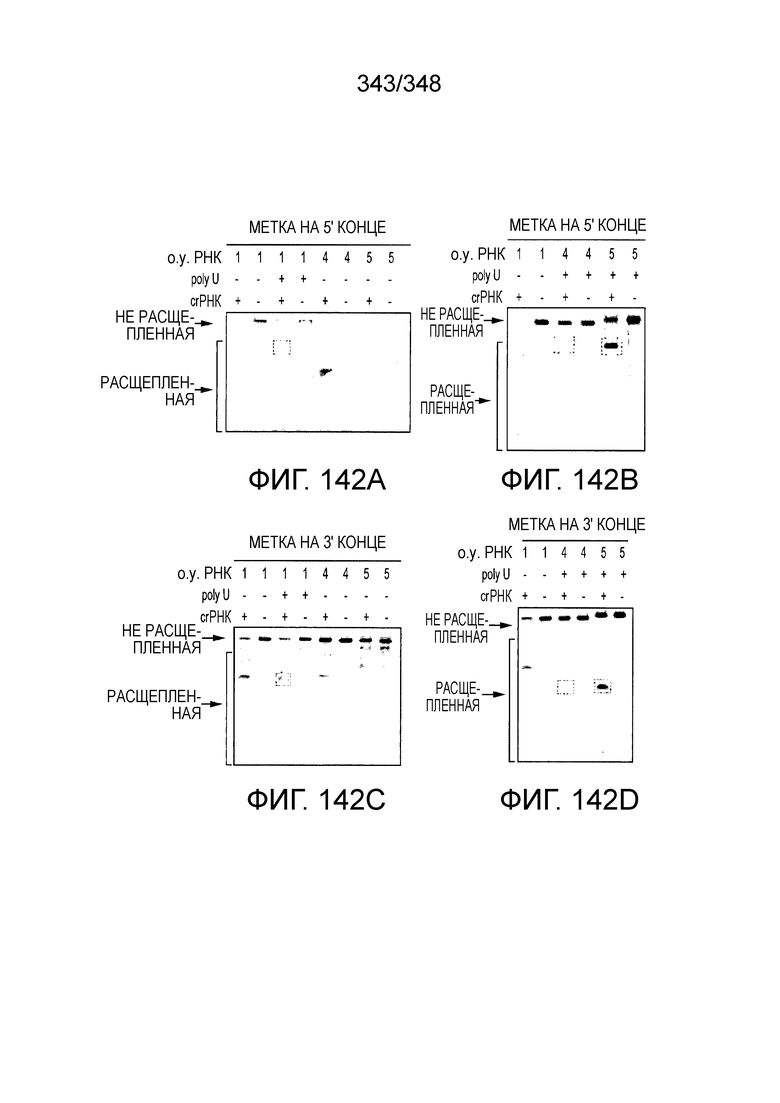
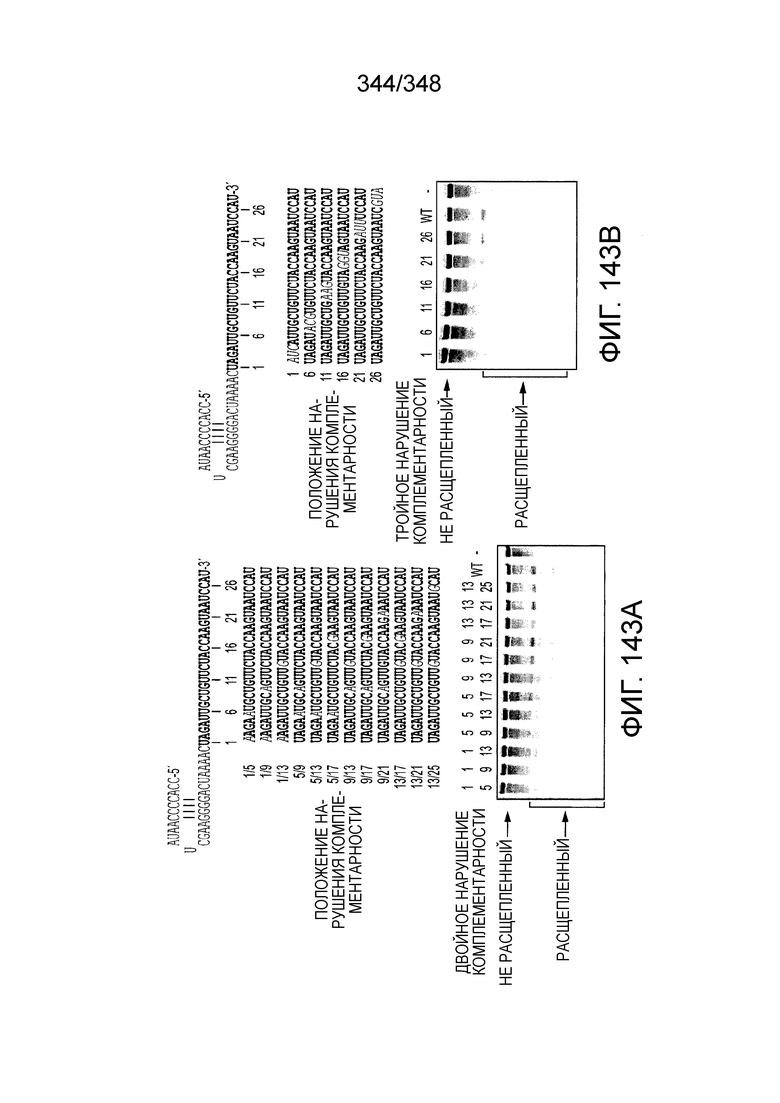
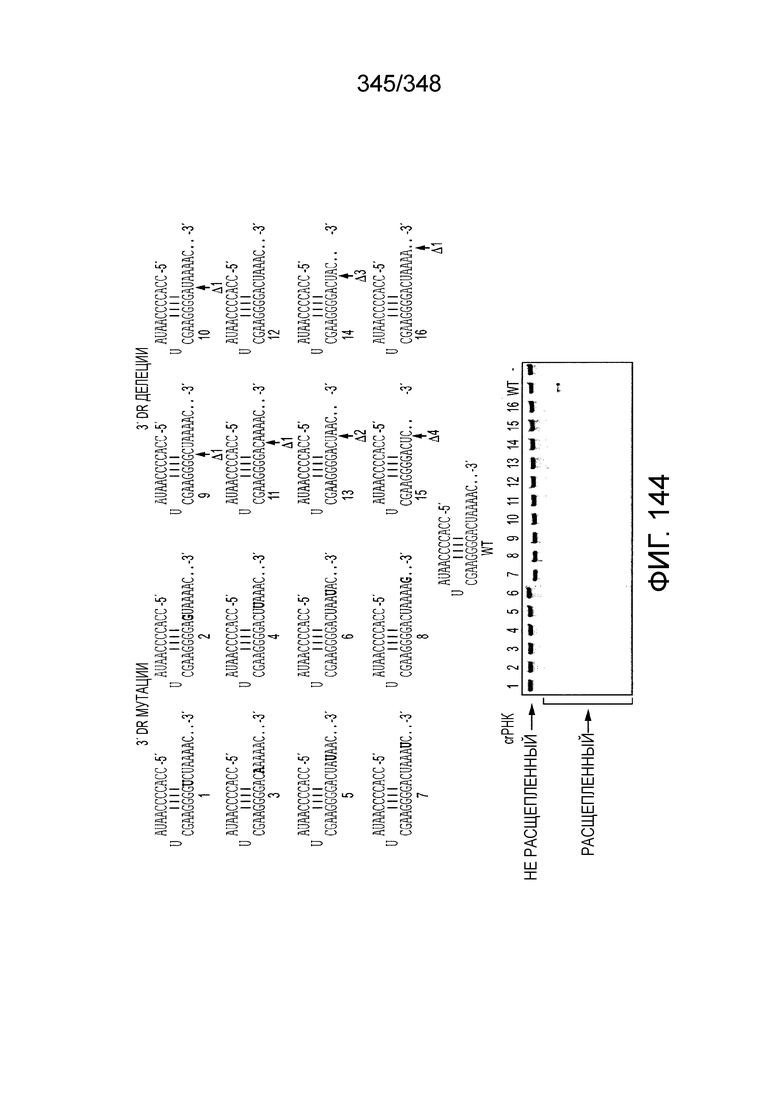
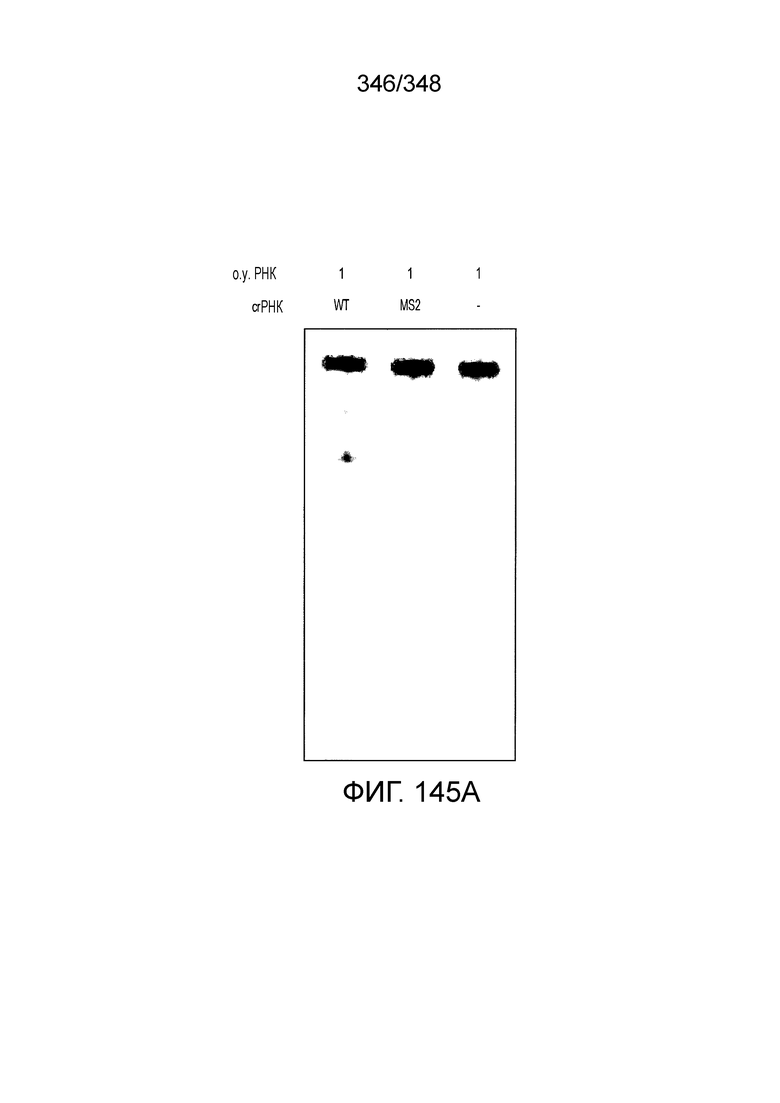
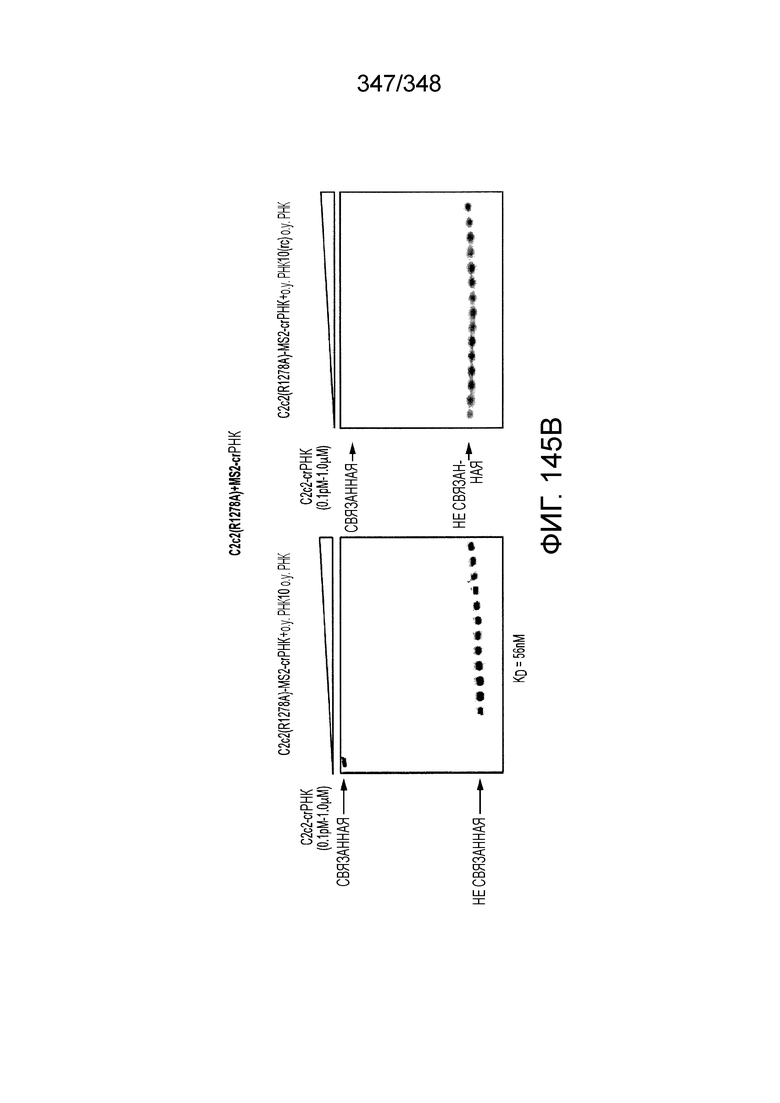
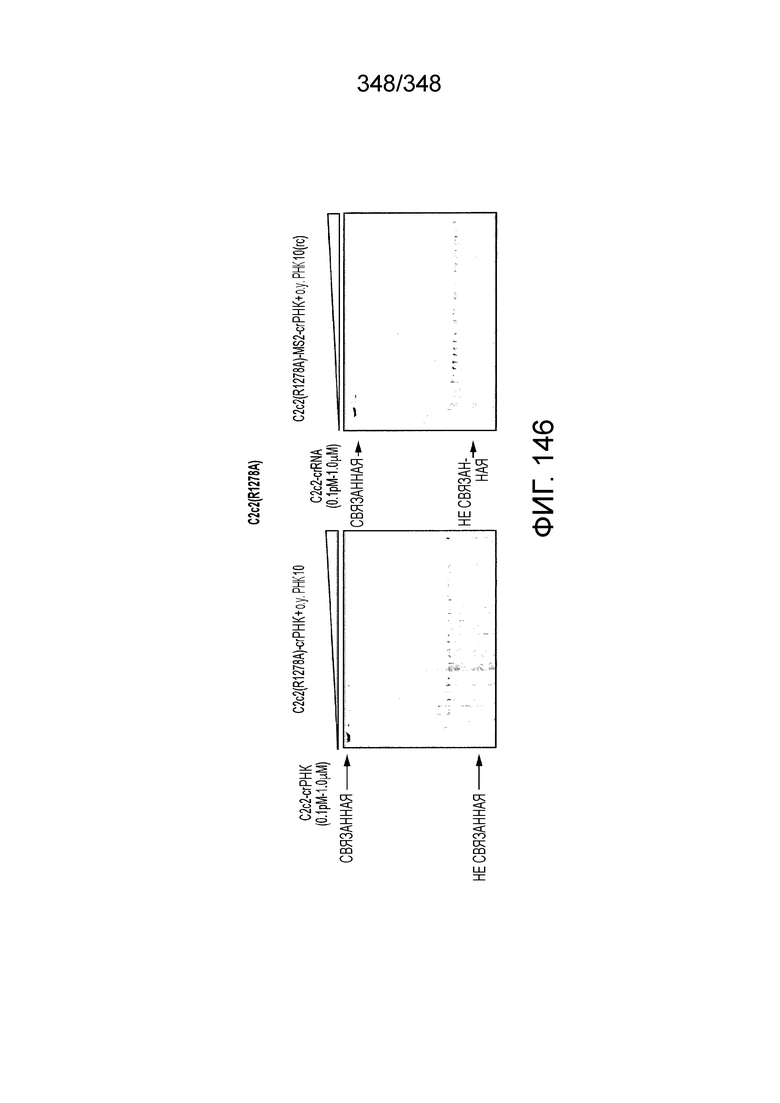
Изобретение относится к области биотехнологии, в частности к способу модификации представляющего интерес локуса-мишени, включающему доставку в указанный локус не встречающейся в природе или сконструированной композиции, содержащей эффекторный белок CRISPR-Cas типа VI класса 2, содержащий 2 домена HEPN, и один или более компонентов, являющихся нуклеиновыми кислотами. Также раскрыта не встречающаяся в природе или сконструированная композиция для модификации представляющей интерес РНК, а также содержащая ее система доставки для модификации представляющего интерес локуса-мишени и векторная система для модификации представляющего интерес локуса-мишени, включающая один или более векторов. Изобретение также относится к клетке для модификации представляющей интерес РНК, а также к содержащей ее клеточной линии. Изобретение эффективно для модификации представляющего интерес локуса-мишени. 9 н. и 51 з.п. ф-лы, 146 ил., 12 табл., 9 пр.
1. Способ модификации представляющего интерес локуса-мишени, включающий доставку в указанный локус не встречающейся в природе или сконструированной композиции, содержащей эффекторный белок CRISPR-Cas типа VI класса 2, содержащий 2 домена HEPN, и один или более компонентов, являющихся нуклеиновыми кислотами, где эффекторный белок образует комплекс с одним или более компонентами, являющимися нуклеиновыми кислотами, один или более компонентов, являющихся нуклеиновыми кислотами, направляют комплекс к представляющему интерес локусу-мишени и комплекс связывается с представляющим интерес локусом-мишенью, где локус-мишень представляет собой РНК, и способ модификации не включает модификацию генетической целостности клеток зародышевой линии человека.
2. Способ по п. 1, в котором модификация представляющего интерес локуса-мишени включает разрыв нуклеотидной цепи.
3. Способ по п. 1, в котором эффекторный белок связан с одним или более функциональными доменами, и необязательно эффекторный белок содержит одну или более мутаций, необязательно в домене HEPN, таких как R597A, Н602А, R1278A и/или Н1283А, посредством чего комплекс может доставлять эпигенетический модификатор или транскрипционный или трансляционный сигнал активации или подавления.
4. Способ по п. 3, в котором функциональный домен модифицирует транскрипцию или трансляцию локуса-мишени.
5. Способ пп. 1-4, в котором эффекторный белок содержит по меньшей мере один или более сигналов ядерной локализации.
6. Способ по п. 1, в котором представляющий интерес локус-мишень предоставлен посредством молекулы нуклеиновой кислоты in vitro.
7. Способ по п. 1, в котором представляющий интерес локус-мишень предоставлен посредством молекулы нуклеиновой кислоты внутри клетки.
8. Способ по п. 7, в котором клетка является прокариотической клеткой.
9. Способ по п. 7, в котором клетка является эукариотической клеткой.
10. Способ по любому из предшествующих пунктов, в котором компонент(ы), являющийся нуклеиновой кислотой, когда он в комплексе с эффекторным белком, способен осуществлять последовательность-специфическое связывание комплекса с последовательностью-мишенью представляющего интерес локуса-мишени.
11. Способ по любому из предшествующих пунктов, в котором компонент(ы), являющийся нуклеиновой кислотой, содержит двойную последовательность прямого повтора.
12. Способ по любому из предшествующих пунктов, в котором эффекторный белок и компонент(ы), являющийся нуклеиновой кислотой, предоставлены посредством одной или более полинуклеотидных молекул, кодирующих полипептиды и/или компонент(ы), являющийся нуклеиновой кислотой, и где одна или более полинуклеотидных молекул функционально организованы для экспрессии полипептидов и/или компонента(ов), являющегося нуклеиновой кислотой.
13. Способ по п. 12, в котором одна или более полинуклеотидных молекул содержат один или более регуляторных элементов, функционально организованных для экспрессии полипептидов и/или компонента(ов), являющегося нуклеиновой кислотой, где необязательно один или более регуляторных элементов содержат промотор(ы) или индуцибельный промотор(ы).
14. Способ по п. 12, в котором полинуклеотидная последовательность, кодирующая эффекторный белок, является кодоноптимизированной для экспрессии в эукариотической клетке.
15. Способ по п. 12 или 13, в котором одна или более полинуклеотидных молекул находятся в одном или более векторах.
16. Способ по п. 12 или 13, в котором одна или более полинуклеотидных молекул находятся в одном векторе.
17. Способ по п. 15 или 16, в котором один или более векторов включают вирусные векторы.
18. Способ по п. 17, в котором один или более вирусных векторов включают один или более ретровирусных, лентивирусных, аденовирусных, аденоассоциированных вирусных векторов или векторов на основе вируса простого герпеса.
19. Способ по любому из пп. 13, 14, в котором одна или более полинуклеотидных молекул находятся в системе доставки.
20. Способ по п. 15 или 16, в котором один или более векторов находятся в системе доставки.
21. Способ по любому из пп. 1-12, в котором комплекс находится в системе доставки.
22. Способ по любому из предшествующих пунктов, в котором не встречающаяся в природе или сконструированная композиция доставляется посредством носителя для доставки, включающего липосому(ы), частицу(ы), экзосому(ы), микровезикулу(ы), генные пушки или один или более вирусный вектор(ов).
23. Не встречающаяся в природе или сконструированная композиция для модификации представляющей интерес РНК, содержащая эффекторный белок CRISPR-Cas типа VI класса 2, содержащий 2 домена HEPN, и один или более компонентов, являющихся нуклеиновыми кислотами, где эффекторный белок образует комплекс с одним или более компонентами, являющимися нуклеиновыми кислотами, один или более компонентов, являющихся нуклеиновыми кислотами, направляет комплекс к представляющему интерес локусу-мишени и связывается с представляющим интерес локусом-мишенью, где локус-мишень представляет собой РНК, и модификация не включает модификацию генетической целостности клеток зародышевой линии человека.
24. Композиция по п. 23, в которой модификация представляющего интерес локуса-мишени включает разрыв нуклеотидной цепи.
25. Композиция по п. 23, в которой эффекторный белок связан с одним или более функциональными доменами, и необязательно эффекторный белок содержит одну или более мутаций, необязательно в домене HEPN, таких как R597A, Н602А, R1278A и/или Н1283А, посредством чего комплекс может доставлять эпигенетический модификатор или транскрипционный или трансляционный сигнал активации или подавления.
26. Композиция по п. 25, в которой функциональный домен модифицирует транскрипцию или трансляцию локуса-мишени.
27. Композиция по любому из пп. 23-26, в которой эффекторный белок содержит по меньшей мере один или более сигналов ядерной локализации.
28. Композиция по п. 23, в которой представляющий интерес локус-мишень предоставлен посредством молекулы нуклеиновой кислоты in vitro.
29. Композиция по п. 23, в которой представляющий интерес локус-мишень предоставлен посредством молекулы нуклеиновой кислоты внутри клетки.
30. Композиция по п. 29, в которой клетка является прокариотической клеткой.
31. Композиция по п. 29, в которой клетка является эукариотической клеткой.
32. Композиция по любому из пп. 23-31, в которой компонент(ы), являющийся нуклеиновой кислотой, когда он в комплексе с эффекторным белком, способен осуществлять последовательность-специфическое связывание комплекса с последовательностью-мишенью представляющего интерес локуса-мишени.
33. Композиция по любому из пп. 23-31, в которой компонент(ы), являющийся нуклеиновой кислотой, содержит двойную последовательность прямого повтора.
34. Композиция по любому из пп. 23-31, в которой эффекторный белок и компонент(ы), являющийся нуклеиновой кислотой, предоставлены посредством одной или более полинуклеотидных молекул, кодирующих полипептиды и/или компонент(ы), являющийся нуклеиновой кислотой, и где одна или более полинуклеотидных молекул функционально организованы для экспрессии полипептидов и/или компонента(ов), являющегося нуклеиновой кислотой.
35. Композиция по п. 34, в которой одна или более полинуклеотидных молекул содержат один или более регуляторных элементов, функционально организованных для экспрессии полипептидов и/или компонента(ов), являющегося нуклеиновой кислотой, где необязательно один или более регуляторных элементов содержат промотор(ы) или индуцибельный промотор(ы).
36. Композиция по п. 34, в которой полинуклеотидная последовательность, кодирующая эффекторный белок, является кодоноптимизированной для экспрессии в эукариотической клетке.
37. Композиция по п. 34 или 35, в которой одна или более полинуклеотидных молекул находятся в одном или более векторов.
38. Композиция по п. 34 или 35, в которой одна или более полинуклеотидных молекул находятся в одном векторе.
39. Композиция по п. 37 или 38, в которой один или более векторов включают вирусные векторы.
40. Композиция по п. 39, в которой один или более вирусных векторов включают один или более ретровирусных, лентивирусных, аденовирусных, аденоассоциированных вирусных векторов или векторов на основе вируса простого герпеса.
41. Композиция по любому из пп. 34, 35, в которой одна или более полинуклеотидных молекул находятся в системе доставки.
42. Композиция по п. 37 или 38, в которой один или более векторов находятся в системе доставки.
43. Композиция по любому из пп. 23-33, в которой комплекс находится в системе доставки.
44. Композиция по любому из пп. 34, 35, в которой не встречающаяся в природе или сконструированная композиция доставляется посредством носителя для доставки, включающего липосому(ы), частицу(ы), экзосому(ы), микровезикулу(ы), генные пушки или один или более вирусных векторов.
45. Векторная система для модификации представляющего интерес локуса-мишени, включающая один или более векторов, где один или более векторов содержат:
а) первый регуляторный элемент, действующий в эукариотической или прокариотической клетке, функционально связанный по меньшей мере с одной нуклеотидной последовательностью, кодирующей направляющую РНК системы CRISPR-Cas, которая гибридизируется с последовательностью молекулы РНК, являющейся мишенью, кодируемой молекулой ДНК в эукариотической или прокариотической клетке, где молекула ДНК кодирует и эукариотическая или прокариотическая клетка экспрессирует по меньшей мере один продукт гена, и
b) второй регуляторный элемент, действующий в эукариотической или прокариотической клетке, функционально связанной с нуклеотидной последовательностью, кодирующей эффекторный белок CRISPR-Cas типа VI класса 2, содержащий 2 домена HEPN, где компоненты (а) и (b) расположены в одном и том же или разных векторах системы,
посредством чего направляющая РНК нацеливается на и гибридизируется с последовательностью-мишенью и эффекторный белок расщепляет молекулу РНК, вследствие чего экспрессия по меньшей мере одного продукта гена изменяется;
и где эффекторный белок и направляющая РНК не встречаются совместно в природе.
46. Система доставки для модификации представляющего интерес локуса-мишени, сконструированная для доставки композиции по любому из пп. 23-44, содержащая векторную систему, включающую один или более векторов, где один или более векторов содержат:
a) первый регуляторный элемент, действующий в эукариотической или прокариотической клетке, функционально связанный по меньшей мере с одной нуклеотидной последовательностью, кодирующей направляющую РНК системы CRISPR-Cas, которая гибридизируется с последовательностью молекулы РНК, являющейся мишенью, кодируемой молекулой ДНК в эукариотической или прокариотической клетке, где молекула ДНК кодирует и эукариотическая или прокариотическая клетка экспрессирует по меньшей мере один продукт гена, и
b) второй регуляторный элемент, действующий в эукариотической или прокариотической клетке, функционально связанной с нуклеотидной последовательностью, кодирующей эффекторный белок CRISPR-Cas типа VI класса 2, содержащий 2 домена HEPN,
где компоненты (а) и (b) расположены в одном и том же или разных векторах системы,
посредством чего направляющая РНК нацеливается на и гибридизируется с последовательностью-мишенью и эффекторный белок расщепляет молекулу РНК, вследствие чего экспрессия по меньшей мере одного продукта гена изменяется;
и где эффекторный белок и направляющая РНК не встречаются совместно в природе.
47. Не встречающаяся в природе или сконструированная композиция по любому из пп. 23-44, векторная система по п. 45 или система доставки по п. 46 для использования в терапевтическом способе лечения.
48. Клетка, не являющаяся эмбриональной клеткой человека, сконструированная так, чтобы содержать или экспрессировать, необязательно индуцируемо или конститутивно, композицию по п. 23 для модификации представляющей интерес РНК, в которой эффекторный белок CRISPR-Cas типа VI класса 2, содержащий 2 домена HEPN, и компонент(ы), являющийся нуклеиновой кислотой, представлены посредством одной или более полинуклеотидных молекул, кодирующих полипептиды и/или компонент(ы), являющийся нуклеиновой кислотой, и где одна или более полинуклеотидных молекул функционально организованы для экспрессии полипептидов и/или компонента(ов), являющегося нуклеиновой кислотой.
49. Клетка по п. 48, в которой одна или более полинуклеотидных молекул содержат один или более регуляторных элементов, функционально организованных для экспрессии полипептидов и/или компонента(ов), являющегося нуклеиновой кислотой, где необязательно один или более регуляторных элементов содержат промотор(ы) или индуцибельный промотор(ы).
50. Клетка по любому из пп. 48, 49, где клетка представляет собой эукариотическую клетку.
51. Клетка по п. 50, где клетка представляет собой клетку млекопитающего.
52. Клетка по любому из пп. 48, 49, где клетка представляет собой прокариотическую клетку.
53. Не встречающаяся в природе или сконструированная композиция по любому из пп. 23-44, векторная система по п. 45 или система доставки по п. 46 для применения в:
- специфической интерференции последовательности РНК,
- специфической регуляции гена последовательности РНК,
- скрининге РНК, или продуктов РНК, или linc-PHK, или некодирующей РНК, или ядерной РНК, или мРНК,
- мутагенезе,
- флуоресцентной in situ гибридизации,
- селекции,
- индукции состояния покоя in vitro или in vivo,
- индукции клеточного цикла in vitro или in vivo,
- снижения роста клеток /или пролиферации in vitro или in vivo,
- индукции клеточной анергии in vitro или in vivo,
- индукции клеточного апоптоза in vitro или in vivo,
- индукции клеточного некроза in vitro или in vivo,
- индукции гибели клеток in vitro или in vivo или
- индукции программируемой гибели клеток in vitro или in vivo.
54. Клеточная линия, состоящая из клеток по любому из пп. 48-52, сконструированных так, чтобы содержать или экспрессировать, необязательно индуцируемо или конститутивно, композицию по любому из пп. 23-33 для модификации представляющей интерес РНК, где клетка не является эмбриональной клеткой человека.
55. Способ мутагенеза, включающий способ модификации представляющего интерес локуса-мишени по любому из пп. 1-22.
56. Способ по п. 55, в котором способ мутагенеза представляет собой способ, основанный на PHK-i или флуоресцентной гибридизации in situ.
57. Не встречающаяся в природе или сконструированная композиция по любому из пп. 23-44, векторная система по п. 45 или система доставки по п. 46 для использования в:
- специфической интерференции последовательности РНК,
- специфической регуляции гена последовательности РНК,
- скрининге РНК, или продуктов РНК, или linc-PHK, или некодирующей РНК, или ядерной РНК, или мРНК,
- мутагенезе,
- флуоресцентной in situ гибридизации,
- селекции,
- индукции состояния покоя in vitro или in vivo,
- индукции клеточного цикла in vitro или in vivo,
- снижения роста клеток /или пролиферации in vitro или in vivo,
- индукции клеточной анергии in vitro или in vivo,
- индукции клеточного апоптоза in vitro или in vivo,
- индукции клеточного некроза in vitro или in vivo,
- индукции гибели клеток in vitro или in vivo или
- индукции программируемой гибели клеток in vitro или in vivo.
58. Способ по любому из пп. 1-22, где указанный способ приводит к:
- специфической интерференции последовательности РНК,
- специфической регуляции гена последовательности РНК,
- скринингу РНК, или продуктов РНК, или linc-PHK, или некодирующей РНК, или ядерной РНК, или мРНК,
- мутагенезу,
- флуоресцентной in situ гибридизации,
- селекции,
- индукции состояния покоя in vitro или in vivo,
- индукции клеточного цикла in vitro или in vivo,
- снижению роста клеток /или пролиферации in vitro или in vivo,
- индукции клеточной анергии in vitro или in vivo,
- индукции клеточного апоптоза in vitro или in vivo,
- индукции клеточного некроза in vitro или in vivo,
- индукции гибели клеток in vitro или in vivo или
- индукции программируемой гибели клеток in vitro или in vivo.
59. Сконструированная, не встречающаяся в природе система CRISPR-Cas для модификации представляющей интерес РНК, содержащая один или более векторов, содержащих:
а) первый регуляторный элемент, действующий в эукариотической или прокариотической клетке, функционально связанный по меньшей мере с одной нуклеотидной последовательностью, кодирующей направляющую РНК системы CRISPR-Cas, которая гибридизируется с последовательностью молекулы РНК, являющейся мишенью, кодируемой молекулой ДНК в эукариотической или прокариотической клетке, где молекула ДНК кодирует и эукариотическая или прокариотическая клетка экспрессирует по меньшей мере один продукт гена, и
b) второй регуляторный элемент, действующий в эукариотической или прокариотической клетке, функционально связанной с нуклеотидной последовательностью, кодирующей эффекторный белок CRISPR-Cas типа VI класса 2, содержащий 2 домена HEPN, где компоненты (а) и (b) расположены в одном и том же или разных векторах системы,
посредством чего направляющая РНК нацеливается на и гибридизируется с последовательностью-мишенью и эффекторный белок расщепляет молекулу РНК, вследствие чего экспрессия по меньшей мере одного продукта гена изменяется;
и где эффекторный белок и направляющая РНК не встречаются совместно в природе.
60. Ассоциированная с CRISPR-Cas векторная система для модификации представляющей интерес РНК, содержащая один или более векторов, содержащих:
a) первый регуляторный элемент, действующий в эукариотической или прокариотической клетке, функционально связанный по меньшей мере с одной нуклеотидной последовательностью, кодирующей направляющую РНК системы CRISPR-Cas, которая гибридизируется с последовательностью молекулы РНК, являющейся мишенью, кодируемой молекулой ДНК в эукариотической или прокариотической клетке, где молекула ДНК кодирует и эукариотическая или прокариотическая клетка экспрессирует по меньшей мере один продукт гена, и
b) второй регуляторный элемент, действующий в эукариотической или прокариотической клетке, функционально связанной с нуклеотидной последовательностью, кодирующей эффекторный белок CRISPR-Cas типа VI класса 2, содержащий 2 домена HEPN,
где компоненты (а) и (b) расположены в одном и том же или разных векторах системы,
посредством чего направляющая РНК нацеливается на и гибридизируется с последовательностью-мишенью и эффекторный белок расщепляет молекулу РНК, вследствие чего экспрессия по меньшей мере одного продукта гена изменяется;
и где эффекторный белок и направляющая РНК не встречаются совместно в природе.
| WO 2015071474 A2, 21.05.2015 | |||
| US 2015079680 A1, 19.03.2015 | |||
| MAKAROVA KIRA S | |||
| et al | |||
| Evolution and classification of the CRISPR-Cas systems, Nature Reviews Microbiology, 2011, 9(6): 467-477 | |||
| WO 2014204724 A1, 24.12.2014 | |||
| US 2010076057 A1, 25.03.2010 | |||
| ПУГАЧ К.С | |||
| и др | |||
| CRISPR-системы адаптивного иммунитета прокариот, Молекулярная биология, 2012, |
Авторы
Даты
2022-05-12—Публикация
2016-06-17—Подача