Область техники настоящего изобретения
Изобретение относится к области генной инженерии, молекулярной биологии и представляет собой способ получения штамма генномодифицированного организма, продуцирующего каталитически активную рекомбиназу Flpe.
Работа выполнена при финансовой поддержке Министерства Науки и Высшего образования Российской Федерации в рамках Соглашения № 075-15-2019-1661 от 31.10.2019.
Предшествующий уровень техники настоящего изобретения
Системы сайт-специфической рекомбинации из бактерий, фагов и дрожжей применяются для генетического анализа модельных организмов, таких как плодовая мушка, мыши (Branda and Dymecki, 2004). Наряду с системой рекомбинации Cre-LoxP из фага P1, наиболее часто используется система рекомбинации Flp-FRT из дрожжей Saсcharomyces cerevisiae. Система Flp-FRT кодируется 2 µm плазмидой (Broach et al., 1982) и состоит из тирозиновой рекомбиназы Flp (Flippase), узнающей сайты рекомбинации, длиной 34 п.н., называемые FRT (Flippase recognition target). Сайт FRT дикого типа, GAAGTTCCTATTCtctagaaaGTATAGGAACTTC, состоит из двух инвертированных повторов длиной 13 п.н. (показаны заглавным шрифтом), разделенных ассиметричным спейсером длиной 8 п.н. (показан строчным шрифтом). Нуклеотидная последовательность спейсера определяет ориентацию сайта FRT в геноме или генетических конструкциях. В природе функция системы Flp-FRT заключается в поддержании копийности 2 µm плазмиды (Chan et al., 2013).
На ранних этапах развития технологии сайт-специфических рекомбиназ у млекопитающих чаще использовалась система Cre-LoxP в силу того, что, по сравнению с Flp, рекомбиназа Cre более активна в организмах или клетках млекопитающих, чья нормальная температура находится в районе +37°С. Эти различия обусловлены тем, что Cre происходит из фага P1, инфицирующего клетки E.coli, обитающие в кишечнике теплокровных животных, а рекомбиназа Flp – из дрожжей, для которых нормальная температура роста около +30°С. Было показано, что Flp термолабильна при температурах около +37-39°С, а максимум активности in vitro проявляет при +23-34°С (Buchholz et al., 1996). С помощью метода молекулярной эволюции была получена термостабильная версия белка Flp, названная Flpe, которая содержит четыре аминокислотных замены P2S, L33S, Y108N и S294P и обладает существенно повышенной активностью при +37°С (Buchholz et al., 1998). Для дальнейшего повышения эффективности работы системы Flp-FRT в клетках млекопитающих была получена версия Flpe с оптимизированным кодонным составом для эффективной трансляции белка в организме домовой мыши, названная Flpo. Уровень активности Flpo был сходен с таковым для Cre (Raymond and Soriano, 2007). Таким образом, с точки зрения аминокислотного состава белки Flpe и Flpo идентичны, однако кДНК Flpe имеет кодонный состав, характерный для дрожжей, а Flpo – для мыши.
Сайт-специфическая рекомбинация представляет наибольший интерес для геномной инженерии и генетического анализа модельных организмов, прежде всего млекопитающих. Стратегия анализа заключается в модификации генома организма путем введения сайтов FRT, в отсутствии рекомбиназы Flp. Далее, тем или иным способом в модифицированные организм или клетки вносится рекомбиназа Flp, которая катализирует сайт-специфическую рекомбинацию по сайтам FRT. С помощью Flp, в зависимости от количества и ориентации сайтов FRT, можно осуществлять интеграцию или удаление фрагмента ДНК из генома, его инверсию, или индуцировать транслокации. В частности, кондиционный нокаут генов у модельных организмов часто реализуют путем фланкирования одного из критически важных экзонов гена двумя сайтами узнавания для сайт-специфических рекомбиназ (FRT или LoxP) с последующим вырезанием этого экзона при экспрессии рекомбиназы (Branda and Dymecki, 2004).
Для реализации технологии кондиционного нокаута у мышей получают так называемые «драйверные» линии трансгенных мышей, экспрессирующие рекомбиназы Cre или Flp под контролем различных вариантов промоторов, которые могут быть конститутивными, тканеспецифическими или индуцибельными. В частности, были получены «драйверные» линии мышей, экспрессирующие Flp, Flpe и Flpo (Dymecki, 1996; Kanki et al., 2006; Kranz et al., 2010; Raymond and Soriano, 2010; Rodríguez et al.; Wu et al., 2009). После скрещивания с линией животных, несущих в геноме сайты FRT, в результате действия рекомбиназы происходит соответствующая модификация генома, в зависимости от количества и взаимной ориентации сайтов FRT.
Кроме того, на основе системы сайт-специфической рекомбинации Flp-FRT был разработан метод геномной инженерии, называемый recombinase mediated cassette exchange, (RMCE, опосредованный-рекомбиназой обмен кассет) (Schlake and Bode, 1994). Практическая реализация RMCE основана на том, что некоторые нуклеотидные замены в последовательностях спейсеров в сайтах FRT не влияют на эффективность рекомбинации. Однако между собой эффективно рекомбинировать могут только сайты FRT с идентичными спейсерами. Использование двух FRT сайтов с различающимся спейсерами позволяет строго сохранять ориентацию фрагмента ДНК после рекомбинации, и тем самым осуществлять точную замену последовательности ДНК в геноме, фланкированной двумя сайтами FRT с различными спейсерами, на внесенную в клетку последовательность ДНК, фланкированную сайтами FRT с теми же спейсерами (Turan et al., 2011; Turan et al., 2013).
Появление технологии геномного редактирования с помощью системы CRISPR/Cas9 позволило существенно упростить и ускорить процесс получения трансгенных животных (Low et al., 2016; Singh et al., 2015), что позволяет эффективно получать новые линии мышей с заданной тканеспецифичностью экспрессии рекомбиназы Flp, а также продуцировать линии животных, несущие в геноме сайты FRT, для создания кондиционного нокаута или проведения RMCE. Это определяет важность пересмотра и оптимизации методов генетического анализа с использованием сайт-специфической рекомбинации. В настоящий момент, все более широкое применение находят, так называемые, технологии редактирования генома без использования ДНК (DNA free genome editing), что прежде всего подразумевает доставку в клетки систем редактирования генома в виде рибонуклеопротеинового комплекса белка Cas9 и гидовой РНК (РНП), а не в виде экспрессионных плазмид. Использование РНП более эффективно, снижает риск модификации нецелевых локусов генома и элиминирует возможность случайной интеграции плазмидной ДНК в нецелевой сайт генома (Metje-Sprink et al., 2019). Концепцию редактирования генома без ДНК можно экстраполировать и на системы сайт-специфической рекомбинации. В частности, рекомбиназы, могут быть доставлены в клетки в виде экспрессионной плазмиды, мРНК или белка. В силу технической простоты и доступности чаще всего для этой цели используют плазмидные конструкции. Однако плазмидная ДНК может интегрировать в случайное место генома, что требует дополнительной проверки клеточной линии или модельного организма на предмет интеграции плазмиды. Использование мРНК сайт-специфических рекомбиназ достаточно эффективно (Ponsaerts et al., 2004), однако при внесении мРНК требуется время для транскрипции и трансляции белка, что снижает эффективность рекомбинации и в случае микроинъекций мРНК в зиготы может способствовать формированию мозаицизма. Учитывая важность и широкое распространение систем сайт-специфической рекомбинации, в том числе Flp-FRT, для манипуляций с геномами модельных организмов, актуальной представляется разработка подходов, основанных на использовании рекомбинантного белка Flp.
До настоящего момента применение рекомбиназы Flp в виде белка не получило широкого распространения, что, вероятно, обусловлено сложностями при экспрессии и очистке Flp из бактериальных систем (Babineau et al., 1985; Meyer-Leon et al., 1987; Pan et al., 1991), а также сниженной активностью Flp и Flpe по сравнению с Cre (Buchholz et al., 1998; Buchholz et al., 1996). В одном из вариантов в бактериальной системе экспрессии получали рекомбинантный белок Flp, слитый с TAT-пептидом, и способный проникать в клетки без использования специальных реагентов для трансфекции белков. TAT-Flp эффективно индуцировал рекомбинацию на модели культур клеток млекопитающих, в том числе эмбриональных стволовых клеток мыши (Patsch et al., 2010). Белок Tat-Flp в настоящий момент коммерчески доступен от компании Excellgen (https://www.excellgen.com/stem-cell-reagents-c-13/flp-recombinase-tatflp-tatnlsflp-htnflp-p-56572.html). Однако использование рекомбинантного белка Flp для геномной инженерии ограничивается лишь этим примером.
Очистка белка Flp
Первые попытки очистить Flp используя бактериальную систему экспрессии не позволяли получить высокоочищенный белок Flp. В частности, трехcтадийная хроматография на анионообменной DEAE-целлюлозе и катионообменном носителе Bio-Rex 70 позволяла получить белок с чистотой около 50% (Babineau et al., 1985).
Модификация процедуры очистки Flp и использование ДНК-аффинной хроматографии позволило получить белок с чистой до 85%. В частности, очистка включала осаждение белков сульфатом аммония из тотального клеточного лизата, обессоливание лизата на колонке Biogel- P6DG, катионообменную хроматографию на колонке Bio-Rex 70, хроматографию на гепариновой колонке и наконец, аффинную хроматографию на сефарозе с ковалентно иммобилизованной ДНК, несущей сайты связывания для Flp (Meyer-Leon et al., 1987). Авторам удалось получить относительно чистый белок и показать его активность в in vitro тесте. Однако описанная процедура очистка занимает четыре дня и достаточно трудоемкая.
Дальнейшее усовершенствование процедуры очистки Flp позволило получить белок из бактериальной системы экспрессии под контролем T7 промотора с чистотой до 98% (Pan et al., 1991). В частности, активный белок Flp удавалось получить при индукции экспрессии при +22°С, но не при +37°С. Протокол очистки включал хроматографию на слабом катионообменном носителе Bio-Rex 70 (Bio-Rad), осаждение белка сульфатом аммония, последующую гель-фильтрацию на носителе Sephacryl S300, и наконец финальную очистку на катионообменном носителе Mono S.
В работе (Buchholz et al., 1998) белок Flpe очищали с помощью трехстадийной хроматографии, используя P11 фосфоцеллюлозную хроматографию (часто применявшуюся для очистки ДНК-модифицирующих ферментов (Lu and Erickson, 1997)), гель-фильтрацию на носителе Sephacryl S20 и катионообменную хроматографию на Mono S носителе. Очищенный белок имел чистоту более 95% по ДСН-ПААГ и использовался в in vitro анализе рекомбинации с использованием линеаризованного плазмидного субстрата с двумя сайтами FRT.
В конечном итоге, модификации методов очистки Flp и Flpe позволили получать белки в количествах, достаточных для кристаллизации и последующего рентгеноструктурного анализа. В работе Chen и коллег использовали модифицированный протокол из работы (Pan et al., 1991). В частности, перед этапом финальной очистки на катионообменной колонке Mono S была добавлена стадия аффинной очистки на ДНК-колонке с иммобилизованными сайтами FRT (Chen et al., 2000). В другой работе использовали систему экспрессии компании Novagen в которой белок Flpe продуцировали с С-концевым 6-гистидиновым эпитопом в векторе pET24d в клетках BL21(DE3)pLysS. Белок очищали с помощью метал-аффинной хелатной хроматографии на колонке Talon (Invitrogen) и затем на аффинной ДНК-колонке с иммобилизованными сайтами FRT. Полученный белок концентрировали на колонках Centricon до концентрации 10 мг/мл (Conway et al., 2003), что позволило получить кристаллы белка Flpe в комплексе с субстратом, пригодные для рентгеноструктурного анализа.
Выход белка Flp в бактериальных системах экспрессии крайне низок вследствие наличия в кДНК Flp неоптимальных для E.coli кодонов из дрожжей. Поэтому для увеличения продукции Flpe в бактериальной системе проводили оптимизацию кодонов Flpe для экспрессии в E.coli (Jahnz and Schwille, 2005).
В бактериальной системе экспрессии получали белок Flp, слитый с пептидом из TAT белка вируса иммунодефицита человека. Очистка белка осуществлялась с помощью метал-хелатной аффинной хроматографии за N-концевой 6-гистидиновый эпитоп. Полученный белок эффективно проникал в клетки и индуцировал рекомбинацию в репортерной системе (Patsch et al., 2010). Это указывает на то, что добавление N-концевого 6-гистидинового эпитопа и TAT-пептида не нарушают каталитическую активность Flpe. Описанный в данном изобретении рекомбинантный белок 6His-S-Flpe, продуцируемый штаммом Escherichia coli BL21(DE3)/pET32v11-Flpo, также содержит N-концевые 6-гистидиновый, S-эпитопы и NLS и после очистке сохраняет каталитическую активность.
В одной из последних работ получали каталитически активный белок Flp и его версию, слитую с белком SUMO, в бактериальной системе экспрессии pBAD (Invitrogen). Очистка белка осуществлялась за C-концевой 8-ми гистидиновый эпитоп с помощью метал-хелатной аффинной хроматографии (Ma et al., 2019).
Помимо бактериальных систем экспрессии для продукции белка Flpe использовали бакуловирусную систему экспрессии и FLAG-аффинную очистку (Jensen et al., 2017). Однако, продукция белков в такой системе имеет ряд минусов, таких как высокая стоимость, существенно меньший выход, ограниченная возможность масштабирования за счет дорогостоящей аффинной хроматографии с иммобилизованным FLAG-пептидом и необходимость культивирования больших объемов клеток насекомых. Помимо этого, для экспрессии белка необходимо получение рекомбинантного бакуловируса.
Альтернативным способом получения каталитически активного белка Flp является in vitro трансляция (Amin and Sadowski, 1989). Преимуществом такой системы является получение практически 100% чистого белка, однако это сопряжено с низким выходом белка и высокой стоимостью масштабирования.
Учитывая эти факторы, наиболее оптимальной представляется продукция белка Flpe в бактериальной системе экспрессии.
In vitro анализ активности рекомбиназы Flp
Для оценки активности очищенного белка Flp был разработан in vitro тест на рекомбинацию с использованием в качестве субстрата линеаризованных или кольцевых форм плазмидной ДНК, несущие сайты FRT. При использовании кольцевых плазмид продукты реакции рекомбинации линеаризуют соответствующими эндонуклеазами рестрикции (Babineau et al., 1985; Gates and Cox, 1988; Meyer-Leon et al., 1987). Детекция продуктов рекомбинации, как правило, основана на определении размера и количества продуктов реакции с помощью электрофореза в агарозном геле. Такой подход остается актуальным и в настоящий момент, что подтверждается недавними публикациями (Ma et al., 2019). В других модификациях теста на рекомбинацию использовали линеаризованный фрагмент ДНК, радиоактивно меченный по концам. В этом случае, кольцевой продукт рекомбинации, вырезаемый из субстрата, не имел радиоактивной метки, а линейный продукт рекомбинации был радиоактивно мечен (Buchholz et al., 1998; Buchholz et al., 1996).
Помимо этого, был разработан чувствительный метод оценки эффективности рекомбинации при использовании очищенного белка Flpe на основе двухцветной флуоресцентной кросскорреляционной спектроскопии. В основе метода лежит флуоресцентное мечение линейного субстрата двумя флуорофорами, таким образом, чтобы после вырезания фрагмента ДНК, фланкированного сайтами FRT, каждый из продуктов рекомбинации был мечен лишь одним флуорофором (Jahnz and Schwille, 2005). При подборе условий реакции рекомбинации in vitro авторы использовали Mg2+ в реакционном буфере, установили, что большинство широко используемых детергентов (Tween-20, Triton X-100, Noniodet P-40 и т.д.) ингибируют рекомбиназную активность Flp, а также рекомендовали использовать обработку продуктов реакции протеиназой К для высвобождения ДНК от связанной с ней Flp (Jahnz and Schwille, 2005).
Данное изобретение решает задачу получения бактериального штамма, позволяющего эффективно продуцировать рекомбинантный белок Flpe, слитый с 6-гистидиновым и S-эпитопами (6His-S-Flpe). Описан способ очистки 6His-S-Flpe с помощью катионообменной хроматографии, позволяющий получить каталитически активную рекомбиназу Flpe с выходом около 70 мкг с 1 мл культуры. Белок Flpe может быть использован для геномной инженерии в модельных организмах и клеточных линиях и доставлен в клетки млекопитающих с помощью микроинъекций, электропорации или с использованием коммерчески доступных реагентов для трансфекции белков. Микроинъекции рекомбинантного белка Flp в эмбрионы модельных организмов, несущих в геноме сайты FRT, можно использовать для интеграции фрагментов ДНК в целевое место генома, для удаления геномных последовательностей и получения кондиционного нокаута гена, для проведения RMCE. Инъекции Flp можно рассматривать как альтернативу скрещиванию с «драйверными» линиями животных, экспрессирующими Flp. Применение белка Flpe может повысить эффективность геномной инженерии, расширить арсенал используемых методов, элиминировать риск нецелевой интеграции генетических конструкций для экспрессии Flp в геном клетки-реципиента.
Осуществление/Раскрытие настоящего изобретения
Изобретение решает задачу получения бактериального штамма, эффективно продуцирующего рекомбинантный белок 6His-S-Flpe, представляющий собой сайт-специфическую рекомбиназу Flpe, модифицированную путем добавления N-концевых 6-гистидинового и S-эпитопов, которые могут быть использованной для аффинной очистки рекомбинантного белка (Block et al., 2009; Raines et al., 2000). Помимо этого, продуцируемый в бактериальной системе рекомбинантный белок может быть очищен на основе его физико-химических свойств с помощью катионообменной хроматографии и/или гель-фильтрации. Аминокислотная последовательность рекомбинантного белка содержит сайты расщепления для протеаз тромбина, энтеропептидазы (энтерокиназы) и TEV (протеаза вируса табачной мозаики). При необходимости, с помощью протеолитического расщепления удаляются 6-гистидиновый и S-эпитопы и получается белок Flpe, несущий на N-конце лишь последовательность ядерной локализации (NLS) из большого Т-антигена вируса SV40 (Kalderon et al., 1984). Присутствие NLS значительно повышает сродство белка Flpe к катионообменным носителям, что делает возможным эффективную (более 90% чистоты, Фиг.3) одностадийную очистку данного белка. В другом техническом варианте очищают рекомбинантный белок, не проводя протеолитическое отщепление 6-гистидинового и S-эпитопов, что не влияет на функциональную активность рекомбиназы.
Плазмидный вектор pET32v11-Flpo получают с помощью методов генной инженерии путем амплификации кДНК рекомбиназы Flpo и последующего клонирования в вектор pET32v11, таким образом, чтобы кДНК Flpo с N-концевой NLS находилась в одной рамке считывания с 6-гистидиновым и S-эпитопами и аминокислотными последовательностями сайтов расщепления тромбина, энтерокиназы и протеазы TEV.
Штамм Escherichia coli BL21(DE3)/pET32v11-Flpo, продуцирующий рекомбинантный белок 6His-S-Flpe, получают путем трансформации компетентных клеток штамма Escherichia coli BL21(DE3) плазмидным вектором pET32v11-Flpo. Штамм культивируют на селективной среде, содержащей антибиотик ампициллин в концентрации 50 мкг/мл и хранят при -80°С в среде LB, содержащей 15% глицерина.
Для получения рекомбинантного белка 6His-S-Flpe клетки штамма-продуцента BL21(DE3)/pET32v11-Flpo культивируют в богатой среде TB, индукцию синтеза рекомбинантного белка осуществляют в течение 16 часов при температуре +18°С путем внесения в среду индуктора ИПТГ (изопропил-β-D-1-тиогалактопиранозид) до конечной концентрации 0.8 мМ. Суспензию клеток E.coli разрушают в буфере (50 мМ Трис-HCl pH=7.4, 50 мМ NH4SO4, 1 мМ ЭДТА, 0.5 мМ ДТТ, 1.44 мМ β-меркаптоэтанол, 1 мМ фенилметилсульфонил фторид, 1х кратный коктейль ингибиторов протеаз VII (Calbiochem)), рекомбинантный белок 6His-S-Flpe очищают с помощью катионообменной хроматографии. В других воплощениях метода рекомбинантный белок очищают с помощью (i) гель-фильтрации и катионообменной хроматографии, (ii) метал-хелатной аффинной хроматографии, (iii) метал-хелатной аффинной хроматографии, гель-фильтрации и катионообменной хроматографии.
Идентичность очищенного белка подтверждают с помощью ДСН-ПААГ и Вестерн-блоттинга.
Специфическую активность очищенного рекомбинантного белка 6His-S-Flpe проверяют с помощью in vitro теста на рекомбинацию, используя в качестве субстрата кольцевую плазмидную ДНК, несущую сайты FRT.
Штамм клеток Escherichia coli BL21(DE3)/pET32v11-Flpo депонирован в Биоресурсный Центр Всероссийской коллекции Промышленных Микроорганизмов (БРЦ ВКПМ) НИЦ «Курчатовский институт» - ГосНИИГенетика под номером ВКПМ В-13890.
Краткое описание фигур
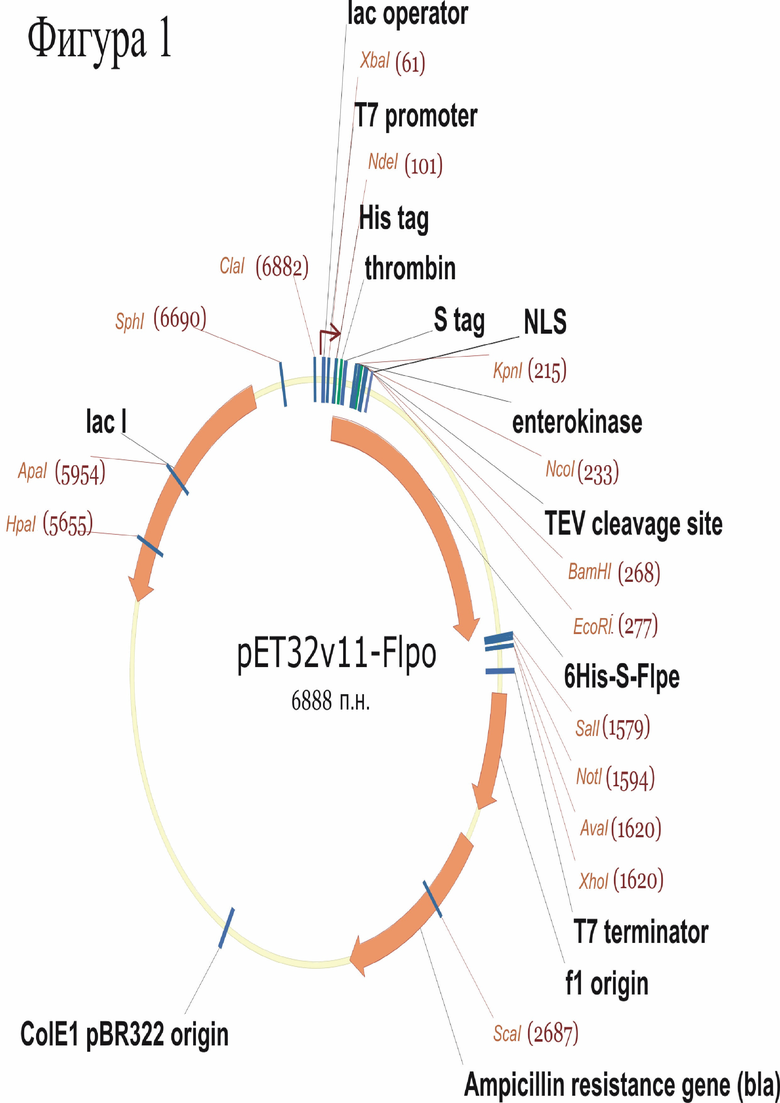
На Фиг.1 показана генетическая карта плазмидной конструкции pET32v11-Flpo, кодирующей рекомбинантный белок 6His-S-Flpe. Показаны основные элементы плазмиды: Ampicillin resistance gene (bla) – ген устойчивости к ампициллину (β-лактамаза); lacI – ген lac репрессора из E.coli; ColE1/pBR322 origin – точка начала репликации; f1 origin – точка начала репликации из бактериофага f1; 6His-S-Flpe – кодирующая последовательность рекомбинантного белка 6His-S-Flpe; lac operator – lac оператор; T7 promoter – T7 промотор; T7 terminator – T7 терминатор; His tag и S-tag – 6-ти гистидиновый и S-эпитопы; thrombin, enterokinase, TEV cleavage site – сайты расщепления соответствующими протеазами. Кроме того, показан ряд уникальных сайтов рестрикции (EcoRI, SalI, BamHI и т.д.).
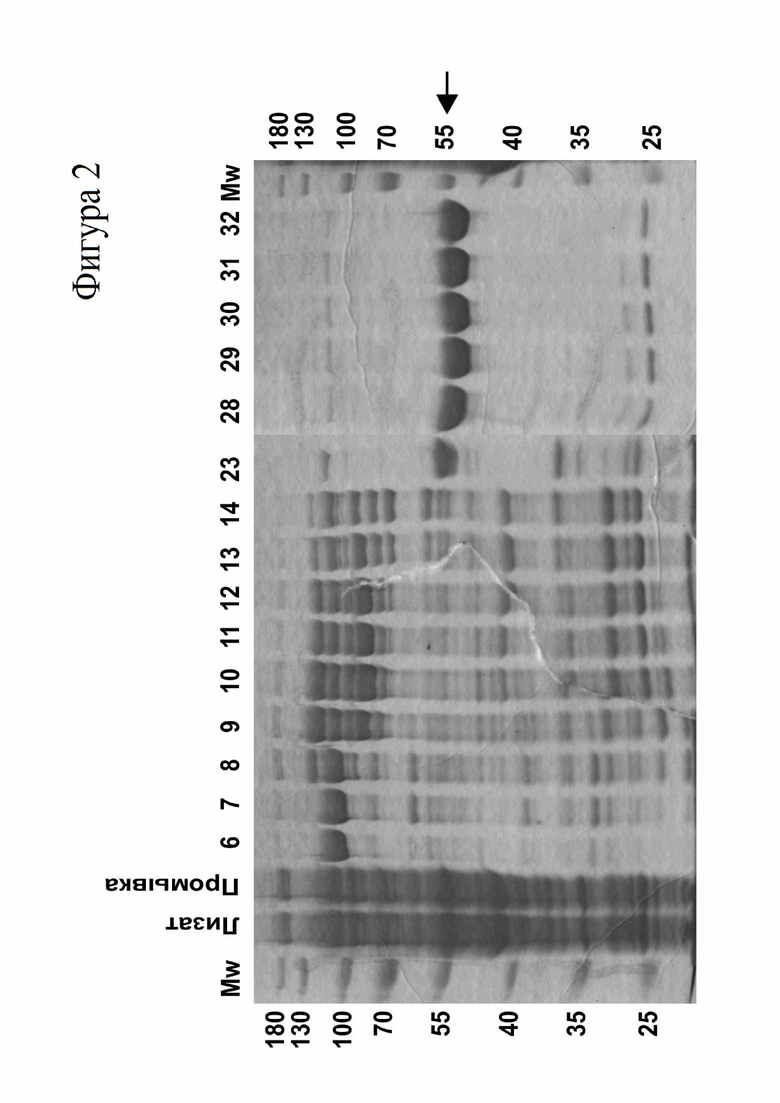
На Фиг.2 показаны результаты анализа с помощью ДСН-ПААГ фракций, элюированных с катионообменной колонки при очистке рекомбинантного белка 6His-S-Flpe. Номера фракций указаны сверху. Лизат: исходный лизат клеток E.coli, нанесенный на колонку. Промывка: фракции, сошедшие при промывке колонки. Mw - маркер молекулярного веса белков: PageRuler™ Prestained Protein Ladder, 10 to 180 kDa (ThermoFisher Scientific). Молекулярный вес полос маркера указан слева и справ от рисунка. Полоса, соответствующая белку 6His-S-Flpe, показана стрелкой.
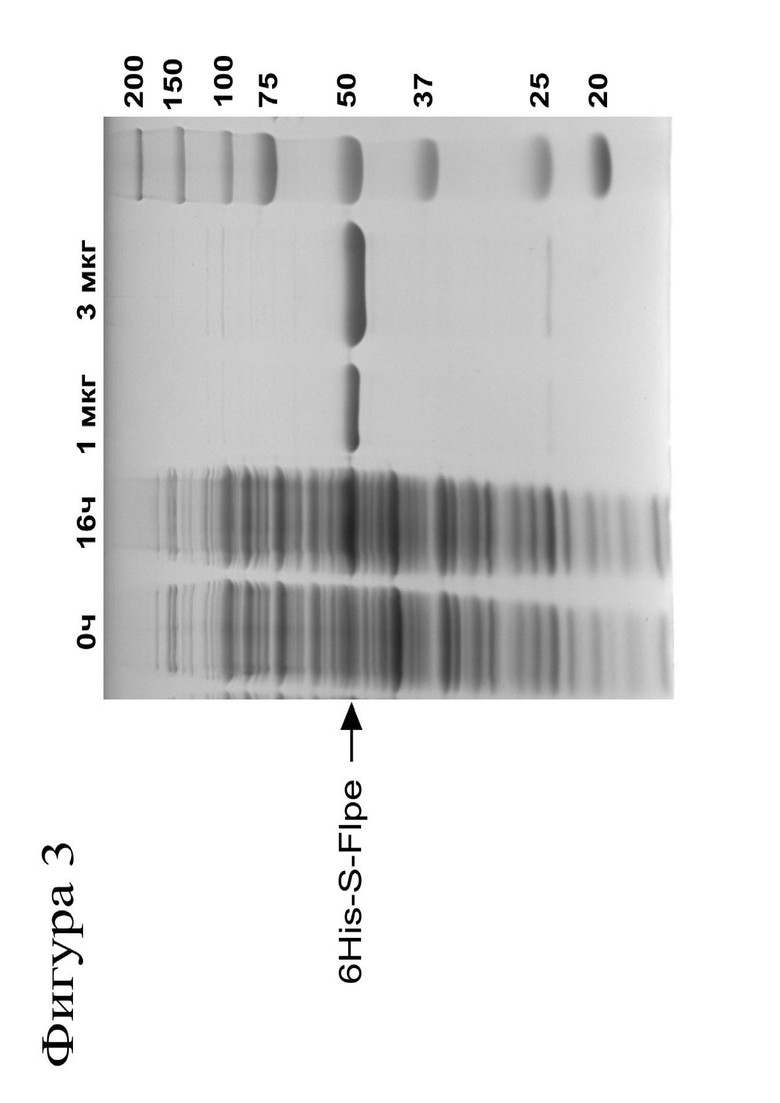
На Фиг.3 показаны результаты анализа с помощью ДСН-ПААГ очищенного рекомбинантного белка 6-His-S-Flpe (дорожки «1 мкг» и «3 мкг») и образцов лизатов клеток штамма E.coli BL21 (DE3)/pET32v11-Flpo до индукции синтеза рекомбинантного белка (дорожка «0ч») и после культивации клеток штамма в среде, содержащий индуктор синтеза белка ИПТГ в концентрации 0.8 мМ в течение 16 часов при +18°С (дорожка «16ч»). Справа указано положение полос маркера молекулярного веса белков Precision Plus Dual Color Protein Standard, (#161-0374 Bio-Rad). Полоса, соответствующая белку 6His-S-Flpe, показана стрелкой.
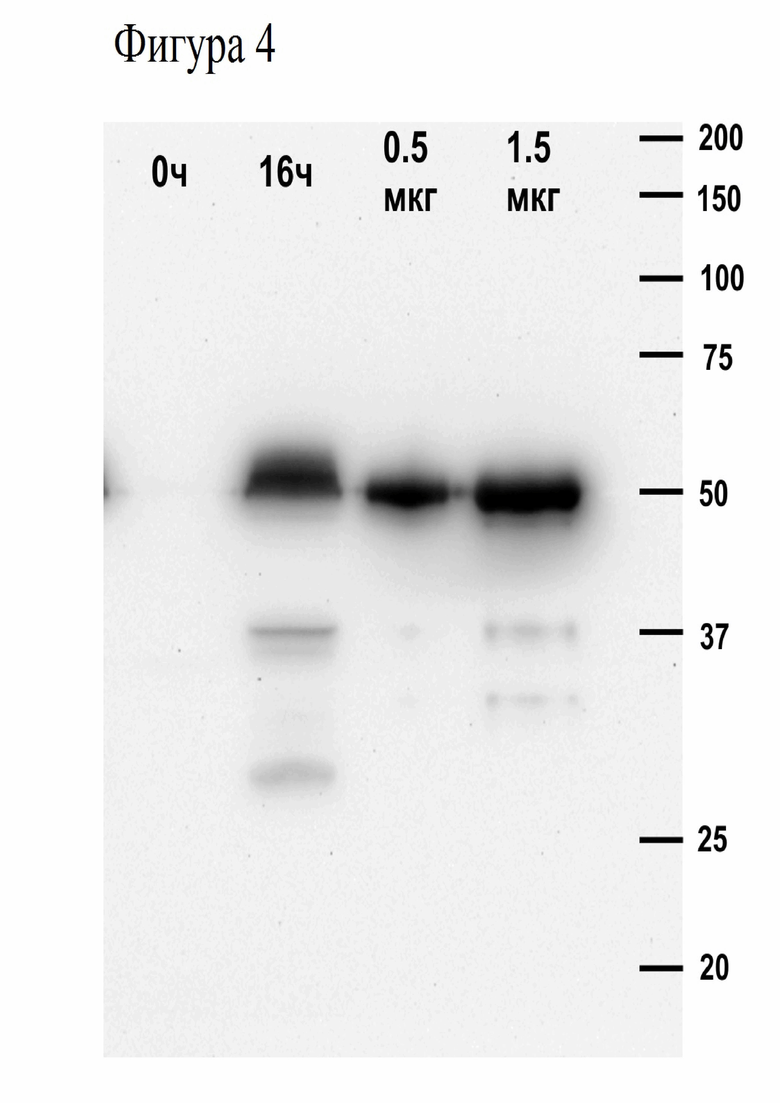
На Фиг.4 показаны результаты Вестерн-блот анализа очищенного рекомбинантного белка 6-His-S-Flpe (дорожки «0.5 мкг» и «1.5 мкг») и образцов лизатов клеток штамма E.coli BL21 (DE3)/pET32v11-Flpo до индукции синтеза рекомбинантного белка (дорожка «0ч») и после культивации клеток штамма в среде, содержащий индуктор ИПТГ в концентрации 0.8 мМ в течение 16 часов при +18°С (дорожка «16ч»). Для Вестерн-блот анализа использовали мышиные моноклональные антитела к 6-гистидиновому эпитопу (Sigma, #H1029, разведение 1:2000). Справа указано положение полос маркера молекулярного веса белков Precision Plus Dual Color Protein Standard, (#161-0374 Bio-Rad).
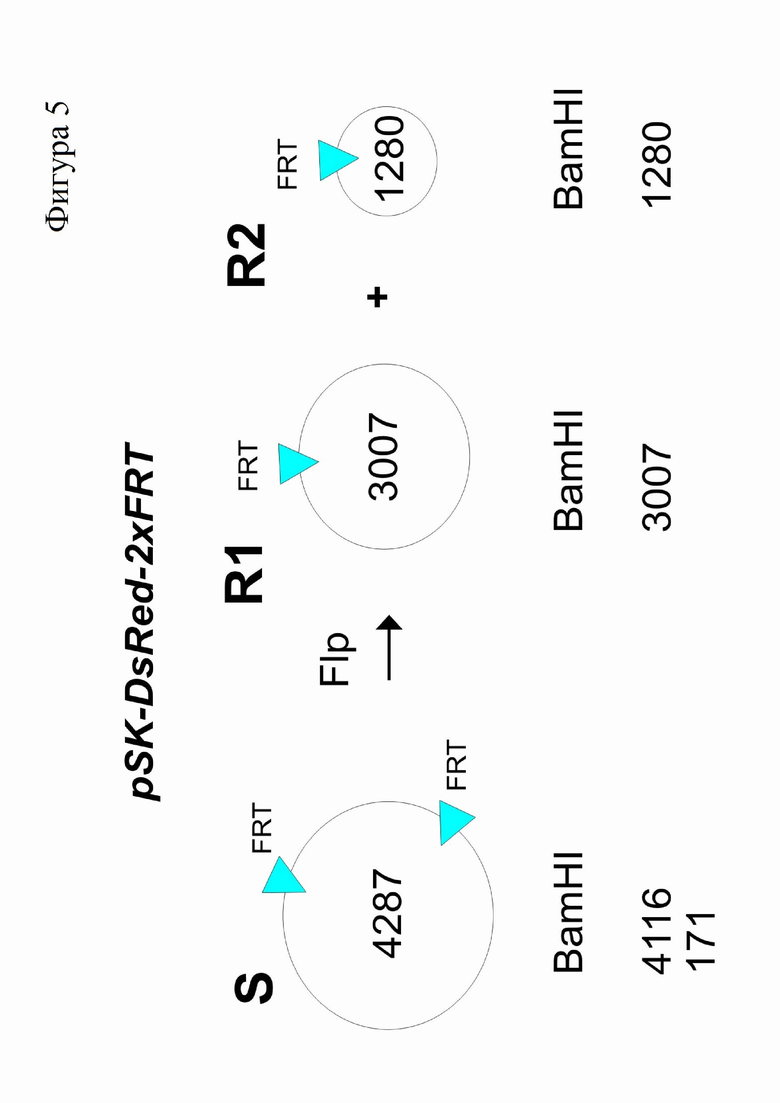
На Фиг.5 показана схема проведения vitro теста на рекомбинацию с использованием модельной плазмиды pSK-DsRed-2xFRT. В результате Flpe-опосредованной рекомбинации из кольцевой плазмиды длиной 4287 п.н. (S) вырезается фрагмент ДНК, фланкированный двумя однонаправленными сайтами FRT. В результате рекомбинации образуются два кольцевых продукта рекомбинации длиной 3007 (R1) и 1280 (R2) п.н. Под схемой приведены размеры линейный фрагментов ДНК, образующихся после обработки субстрата и продуктов реакции эндонуклеазой рестрикции BamHI.
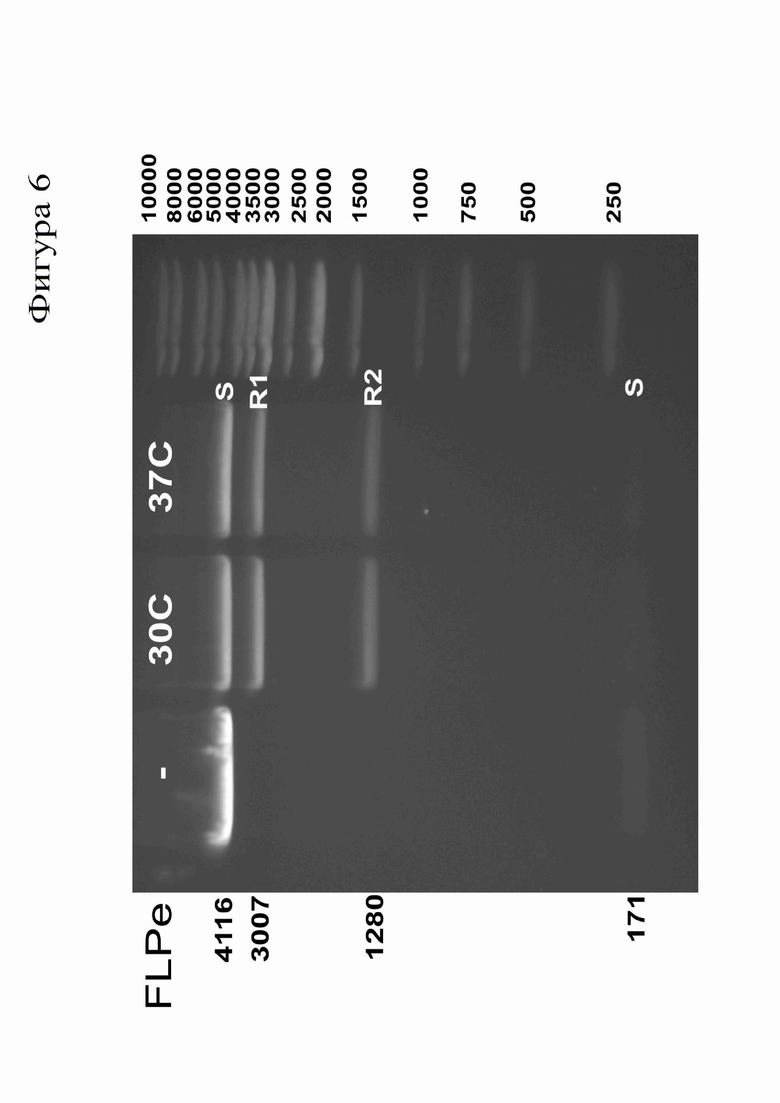
На Фиг.6 показаны результаты in vitro теста на рекомбинацию с использованием модельной плазмиды pSK-DsRed-2xFRT. Продукты реакции рекомбинации анализировали в 1%-ом TAE-агарозном геле. Дорожка «-» – субстрат инкубировали при +37°С без добавления 6His-S-Flpe (Flpe); дорожки «30С» и «37С» – субстрат инкубировали в присутствии 593 нМ 6His-S-Flpe при +30°С и +37°С, соответственно. Размеры продуктов реакции указаны слева. Размеры фрагментов маркера молекулярного веса ДНК (O'GeneRuler 1kb DNA Ladder, ThermoFisher Scientific), показаны справа.
Примеры осуществления настоящего изобретения
Пример 1.
Получение рекомбинантной плазмиды, кодирующей кДНК белка Flpo, слитую с 6-гистидиновым и S-эпитопами
Рекомбинантную плазмиду для продукции в бактериальной системе экспрессии рекомбинантного белка Flpe конструируют с помощью методов генной инженерии. Для этого кДНК рекомбиназы Flpe с оптимизированным кодонным составом для экспрессии в клетках мыши амплифицируют с помощью ПЦР на матрице плазмиды pCAG-Flpo (Addgene #60662, (Xue et al., 2014)) с олигонуклеотидными праймерами 5’ – gaattcgctcctaagaagaagag -3’ и 5’ – gtcgactcagatccgcctgttg-3’, содержащими сайты узнавания эндонуклеаз рестрикции EcoRI и SalI, соответственно (выделены жирным шрифтом). Продукт амплификации анализируют с помощью электрофореза в 1%-ом агарозном геле, очищают из агарозного геля с помощью набора реагентов QiaexII (Qiagen) и клонируют в вектор pCR-Blunt (Invitrogen) получая вектор pCR-Flpo ES. Далее определяют полную нуклеотидную последовательность амплифицированного фрагмента с помощью секвенирования по Сэнгеру.
Далее, на основе вектора pET-32a(+) (Merck) получают модифицированный вектор pET32v11. Для этого, из вектора pGEX-6P1 (GE Healthcare) клонируют фрагмент NotI-XhoI, содержащий стоп-кодоны, в вектор pET-32a(+) по сайтам NotI-XhoI. В полученный вектор по сайтам NcoI-BamHI (выделены жирным шрифтом) клонируют фрагмент ДНК (5- ccatgggcagatctgaaaacctgtactttcagagcggatcc-3), содержащий сайт расщепления протеазой TEV. Далее, из полученного вектора удаляют NdeI-NdeI фрагмент, несущий кодирующую последовательность тиоредоксина, и получают вектор pET32v11 (SEQ ID NO: 1).
В векторе pET32v11 сайт расщепления для протеазы TEV расположен таким образом, чтобы после очистки рекомбинантного белка с помощью иммобилизованной металло-аффинной хроматографии используя протеазу TEV, при необходимости можно было отщепить N-концевой фрагмент очищенного белка, включающий 6-гистидиновый и S-эпитопы.
Далее EcoRI-SalI фрагмент, содержащий кДНК Flpo, клонируют из вектора pCR-Flpo ES в плазмидный вектор pET32v11 по сайтам EcoRI-SalI таким образом, чтобы кДНК Flpo находилась в одной рамке считывания с N-концевым 6-гистидиновым эпитопом. Полученную рекомбинантную плазмиду называют pET32v11-Flpo.
Полученная плазмидная конструкция имеет размер 6888 п.н., (SEQ ID NO: 2). Генетическая карта плазмиды pET32v11-Flpo представлена на Фиг.1. Слитый рекомбинантный белок 6His-S-Flpe, кодируемый данной плазмидой, имеет расчетный молекулярный вес 56.2 кДа и состоит из 491 аминокислоты (SEQ ID NO: 3).
Пример 2.
Процедура получения штамма бактерий E.coli BL21(DE3)/pET32v11-Flpo, продуцирующего рекомбинантный белок 6-His-S-Flpe
Плазмидный вектор pET32v11-Flpo трансформируют в штамм E.coli BL21 (DE3) с помощью стандартной методики трансформации бактерий (Titus, 1991) и высевают на LB агар (0.171 M NaCl, 1% триптон, 0.5% дрожжевой экстракт, 1.5 % агар), содержащий селективный антибиотик ампициллин в концентрации 50 мкг/мл и растят в течение 16 часов при температуре +37°С. Единичные колонии бактерий, выросших на селективной среде, пересевают штрихом на свежую селективную чашку, и растят в течение 16 часов при температуре +37°С. С чашки Петри бактериальной петлей берут единичную колонию бактерий и инокулируют 5 мл жидкой ростовой среды LB, содержащей ампициллин в концентрации 100 мкг/мл и растят в течение 16 часов при температуре +37°С и постоянном перемешивании. 200 мкл ночной культуры переносят в 2 мл среды LB содержащей ампициллин в концентрации 100 мкг/мл и растят в течение 2 часов при температуре +37°С. Клетки E.coli осаждают центрифугированием, удаляют супернатант, осадок ресуспендируют в 500 мкл среды для заморозки (жидкая среда LB, содержащая 15% глицерина). Полученный штамм-продуцент хранят при температуре -80°С.
Пример 3.
Процедура получения рекомбинантного белка 6His-S-Flpe в бактериальной системе экспрессии
Клетки штамма E.coli BL21(DE3) трансформируют плазмидой pET32v11-Flpo и высевают на чашку Петри с LB агаром, содержащим 0.5% глюкозу и 50 мкг/мл ампициллина. С чашки Петри смывают несколько колоний бактерий и ресуспендируют их в прогретой до +37°С среде Terrific Broth (TB; 17 мМ KH2PO4, 72 мМ K2HPO4, 5 мМ MgSO4, 1.2% триптон, 2.4% дрожжевой экстракт, 0.5 % глицерин) и инокулируют две 2-х литровые колбы, содержащие по 500 мл жидкой среды TB и ампициллин в концентрации 50 мкг/мл. Бактерии растят при постоянном перемешивании и температуре +37°С в течение 3-5 часов до достижения оптической плотности 0.4 при длине волны 600 нм. Далее колбы охлаждают до +18°С и индуцируют экспрессию рекомбинантного белка путем добавления ИПТГ до конечной концентрации 0.8 мМ, добавляют ампициллин до концентрации 50 мкг/мл и растят бактерии при температуре +18°С в течение 16 часов при постоянном перемешивании (220 оборотов/мин). На Фиг.3 (дорожка «16ч») показан результат анализа образцов культуры клеток E.coli до и после индукции синтеза рекомбинантного белка. Видно, что в лизате индуцированных клеток накапливается белок с молекулярным весом около 50 кДа (показан стрелкой), который мигрирует на одном уровне с очищенным белком 6His-S-Flpe.
Клетки осаждают центрифугированием при 5000 об/мин и температуре +4°С в течение 20 минут, супернатант удаляют, а осадок промывают с использованием 50 мл фосфатно-солевого буфера (0.01 М натрия фосфат, 0.138 М NaCl, 0.0027 M KCl, pH=7.4), предварительно охлажденного до +4°С, и затем клетки осаждают центрифугированием при 5000 об/мин и температуре +4°С в течение 20 минут. Супернатант удаляют, клеточную массу замораживают в жидком азоте и хранят при -80°С до стадии очистки белка.
Суспензию бактериальных клеток медленно оттаивают на льду, ресуспендируют в 30 мл буфера для лизиса (50 мМ Трис-HCl pH=7.4, 50 мМ NH4SO4, 1 мМ ЭДТА, 0.5 мМ ДТТ, 1.44 мМ β-меркаптоэтанол, 1 мМ фенилметилсульфонил фторид, 1х кратный коктейль ингибиторов протеаз VII (Calbiochem)) и разрушают с помощью гомогенизатора LV1 (Microfluidics).
Полученный лизат бактериальных клеток центрифугируют при 20000 об/мин и температуре +4°С в течение 60 минут, собирают осветленный лизат. Очистку белка проводят на хроматографе среднего давления Acta Pure (GE Healthcare). Осветленный лизат наносят на катионообменную хроматографическую колонку HiSep SP SepFast HighRes объемом 5 мл (Biotoolomics), уравновешенную буфером для лизиса. Колонку промывают дегазированным буфером (50 мМ натрия фосфат pH=6.5, 200 мМ NaCl, 1 мМ ЭДТА, 0.5 мМ ДТТ, 15% глицерин). Связавшиеся с колонкой белки элюируют с помощью градиента соли от 0M NaCl до 1.5M NaCl. Собирают фракции объемом 1 мл. Фракции с максимальным пиком поглощения при длине волны 280 нм анализируют с помощью ДСН-ПААГ в 10 % акриламидном геле (Фиг.2) как описано (Laemmli, 1970). Рекомбинантный белок 6His-S-Flpe эффективно элюируется с колонки при 1.125 M NaCl. Результат анализа фракций, собранных в процессе очистки рекомбинантного белка 6His-S-Flpe, приведен на Фиг.2. Видно, что, начиная с фракции 23 с колонки, начинает элюироваться белок с молекулярным весом около 55 кДа.
Фракции, содержащие очищенный рекомбинантный белок 6His-S-Flpe, замораживают в жидком азоте и хранят при -80°С в аликвотах. Для проверки стабильности белка 6His-S-Flpe размораживают аликвоту, проводят функциональный тест на рекомбинацию in vitro (Фиг.5 и 6) и анализируют образец рекомбинантного белка с помощью ДСН-ПААГ и Вестерн-блоттинга (Фиг.3 и 4).
Пример 4.
Процедура определения идентичности и концентрации очищенного рекомбинантного белка 6His-S-Flpe
Концентрацию очищенного рекомбинантного белка 6His-S-Flpe определяют спектрофотометрически, измеряя поглощение образца белка при длине волны 280 нм по сравнению с буфером для элюции на спектрофотометре NanoDrop ND-8000 (ThermoFisher Scientific). Для расчета концентрации белка используют коэффициент молярной экстинкции для белка 6His-S-Flpe, рассчитанный по его аминокислотной последовательности, и составляющий 61620. При этом 1 единица оптической плотности при длине волны 280 нм соответствует концентрации белка в 0.91 мг/мл.
Идентичность очищенного рекомбинантного белка 6His-S-Flpe определяют с помощью ДСН-ПААГ в 10% акриламидном геле и Вестерн-блоттинга. Готовят образцы лизатов бактериальных клеток штамма E.coli BL21 (DE3)/pET32v11-Flpo до индукции синтеза рекомбинантного белка (0ч) и после культивации клеток штамма в среде, содержащий индуктор ИПТГ в концентрации 0.8 мМ в течение 16 часов при +18°С (16ч). Хранящийся при -70°С очищенный рекомбинантный белок 6His-S-Flpe подвергают двум циклам замораживания-оттаивания и готовят образцы, содержащие 0.5, 1, 1.5 и 3 мкг белка 6His-S-Flpe. Проводят электрофорез белков в 10%-ом акриламидном геле и окрашивают гель красителем Кумасси. На Фиг.3 показано, что очищенный рекомбинантный белок 6-His-S-Flpe (дорожки «1 мкг» и «3 мкг») мигрирует в геле на уровне полосы маркера молекулярного веса 50 кДа, что близко к ожидаемому молекулярному весу белка 6His-S-Flpe в 56 кДа. Чистота очищенного белка (дорожки «1 мкг» и «3 мкг») составляет более 90%. Детектируемое в геле количество белка 6-His-S-Flpe и отсутствие в геле интенсивных полос низкого молекулярного веса, которые могли бы указывать на деградацию белка 6-His-S-Flpe, указывают на стабильность полученного рекомбинантного белка при повторном замораживании и оттаивании.
Для подтверждения идентичности очищенного белка проводят Вестерн-блот анализ. Проводят электрофоретическое разделение описанных выше образцов в 10% акриламидном геле, переносят белки из геля на PVDF мембрану (Millipore) c помощью прибора для электропереноса SV20-SDB (Sigma). Мембрану блокируют в буфере TBS-T (50 мМ Трис-HCl pH=8.0, 138 мМ NaCl, 2.7 мМ KCl, 0.05% Tween-20), содержащем 5% сухое обезжиренное молоко (TBS-T5%). Далее инкубируют мембрану в течение 16 часов при +4°С в буфере TBS-T5% с мышиными моноклональными антителами к 6-ти гистидиновому эпитопу (Sigma, #H1029) в разведении 1:2000. Мембрану три раза промывают в буфере TBS-T и инкубируют в течение 1 часа при комнатной температуре со вторичными антителами против иммуноглобулинов мыши, конъюгированных с пероксидазой хрена (GE Healthcare, #NA931V), в разведении 1:5000. Мембрану три раза промывают в буфере TBS-T, детектируют связавшиеся антитела с помощью набора реагентов Immobilon Western Chemiluminescent HRP Substrate (Millipore). Хемилюминесценцию детектируют с помощью системы гель-документации ChemiDoc XRS (Bio-Rad). На Фиг.4 показан результат Вестерн-блот анализа. Видно, что с помощью антител к 6-ти гистидиновому эпитопу детектируются полосы с молекулярным весом около 50 кДа (дорожки «16 ч», «0.5 мкг» и «1.5 мкг»), что соответствует ожидаемому молекулярному весу белка 6His-S-Flpe. Важно отметить, что иммунореактивная полоса ожидаемого молекулярного веса присутствует лишь в образце лизата клеток штамма E.coli BL21 (DE3)/pET32v11-Flpo после индукции синтеза рекомбинантного белка (дорожка «16 ч»), но не в неидуцированном клеточном лизате. Эти результаты подтверждают идентичность очищенного рекомбинантного белка. В дорожках «0.5 мкг» и «1.5 мкг» детектируются полосы с молекулярным весом около 30 и 37 кДа, которые представляют собой либо продукты деградации очищенного белка, либо продукты преждевременной терминации синтеза белка в клетках E.coli. Суммарная интенсивность полос 37 и 30 кДа составляет около 3% от интенсивности полосы, соответствующей полноразмерному белку. Это подтверждает чистоту и стабильность полученного рекомбинантного белка.
Пример 5.
In vitro тест на сайт-специфическую рекомбинацию
Для проведения реакции рекомбинации in vitro в качестве субстрата используют модельную плазмиду pSK-DsRed2xFRT размером 4287 п.н. (SEQ ID NO: 4), в которой кДНК белка DsRed, фланкирована двумя однонаправленными FRT-сайтами. В результате Flp-опосредованной рекомбинации из кольцевого плазмидного субстрата вырезается фрагмент ДНК с образованием двух кольцевых продуктов рекомбинации. Продукты реакции и субстрат линеаризуют, используя эндонуклеазу рестрикции BamHI и анализируют с помощью электрофореза в 1%-ом агарозном геле. При гидролизе субстрата эндонуклеазой рестрикции BamHI образуются фрагменты ДНК длиной 4116 и 177 п.н. (S); при гидролизе продуктов реакции образуются фрагменты длиной 3007 п.н. (R1) и 1280 п.н. (R2). Схема проведения in vitro теста на сайт-специфическую рекомбинацию показана на Фиг.5.
Для проведения реакции рекомбинации готовят смесь объемом 30 мкл, содержащую 10 мМ Трис-HCl pH=7.5, 10 мМ MgCl2, 50 мМ NaCl, 0.1 мг/мл БСА, 500 нг плазмидной ДНК, 593 нМ 6-His-S-Flpe. Реакцию инкубируют при +30°С или +37°С в течение 30 минут. Добавляют 1 мкл эндонуклеазы рестрикции BamHI (10 ед. активности/мкл; «ThermoFisher Scientific») и инкубируют 1 час при +37°С. Добавляют 1 мкл протеиназы K (20 мг/мл, «Sigma») и инкубируют 10 мин при +56°С. Анализируют продукты реакции в 1%-ом агарозном геле.
На Фиг.6 показан результат теста на сайт-специфическую рекомбинацию. При инкубации плазмидной ДНК pSK-DsRed2xFRT с рекомбинантным белком 6-His-S-Flpe при +30°С и +37°С образуются продукты реакции (R1 и R2) ожидаемого размера. При инкубации плазмиды без добавления белка детектируется лишь фрагменты исходного субстрата (S).
Основные характеристики клеток (по данным паспорта штамма)
1. Родовое и видовое название штамма-хозяина (реципиента): Escherichia coli
2. Номер или наименование штамма: BL21(DE3)/pET32v11-Flpo
3. Способ получения штамма: получен трансформацией.
4. Область применения штамма: получение рекомбинантного белка 6His-S-Flpe
5. Продукт, синтезируемый штаммом: белок 6His-S-Flpe
6. Активность (продуктивность) штамма, а также другие производственные показатели: 70 мкг белка на 1 мл среды
7. Способ определения активности штамма с указанием метода: in vitro тест на рекомбинацию
8. Способ, условия и состав сред для длительного хранения штамма: среда LB + 15% глицерина в ампулах на -80°С
9. Способ, условия и состав сред для размножения штамма: среда LB, содержащая 50 мкг/мл ампициллина
10. Группа уровня риска штамма: I
11. Генетические особенности штамма:
а. Генотип штамма хозяина (мутации, делеции, инверсии, наличие плазмид или профагов, устойчивость (чувствительность) к антибиотикам, фагам и т.д., прочие генетические особенности): генотип штамма BL21 (DE3): F– ompT gal dcm lon hsdSB(rB–mB–) λ(DE3 [lacI lacUV5-T7p07 ind1 sam7 nin5]) [malB+]K-12(λS)
б. Описание рекомбинантной плазмиды
Название плазмиды: pET32v11-Flpo
Размер плазмиды pET32v11-Flpo: 6888 пар нуклеотидов
Полная нуклеотидная последовательность плазмиды: SEQ ID NO: 2.
в. Сведения о векторе, на основе которого сконструирована плазмида:
Название вектора; pET-32a(+)
Происхождение вектора и его основных генетических элементов;
Вектор pET-32a(+) содержит:
- ген устойчивости к ампициллину (β-лактамаза)
- ген lac репрессора (lacI) из E.coli
- точку начала репликации из плазмид ColE1/pBR322 из E.coli.
- точку начала репликации из бактериофага f1
- ген rop из E.coli, кодирующий белок, регулирующий копийность плазмиды
- кодирующую последовательность гена тиоредоксина trxA из E.coli
г. Сведения о клонированной ДНК
Видовая принадлежность донорного организма: Saccharomyces cerevisiae
Общие сведения о клонированном фрагменте, размер и включенные в его состав гены; плазмида содержит кДНК гена FLP1 длиной 1296 п.н., кодируемого 2-микронной плазмидой (Genbank Acc.: NC_001398) из дрожжей Saccharomyces cerevisiae
СПИСОК ЛИТЕРАТУРЫ
Amin, A.A., and P.D. Sadowski. 1989. Synthesis of an enzymatically active FLP recombinase in vitro: search for a DNA-binding domain. Mol Cell Biol. 9:1987-1995.
Babineau, D., D. Vetter, B.J. Andrews, R.M. Gronostajski, G.A. Proteau, L.G. Beatty, and P.D. Sadowski. 1985. The FLP protein of the 2-micron plasmid of yeast. Purification of the protein from Escherichia coli cells expressing the cloned FLP gene. J Biol Chem. 260:12313-12319.
Block, H., B. Maertens, A. Spriestersbach, N. Brinker, J. Kubicek, R. Fabis, J. Labahn, and F. Schäfer. 2009. Immobilized-metal affinity chromatography (IMAC): a review. Methods Enzymol. 463:439-473.
Branda, C.S., and S.M. Dymecki. 2004. Talking about a revolution: The impact of site-specific recombinases on genetic analyses in mice. Dev Cell. 6:7-28.
Broach, J.R., V.R. Guarascio, and M. Jayaram. 1982. Recombination within the yeast plasmid 2mu circle is site-specific. Cell. 29:227-234.
Buchholz, F., P.O. Angrand, and A.F. Stewart. 1998. Improved properties of FLP recombinase evolved by cycling mutagenesis. Nat Biotechnol. 16:657-662.
Buchholz, F., L. Ringrose, P.O. Angrand, F. Rossi, and A.F. Stewart. 1996. Different thermostabilities of FLP and Cre recombinases: implications for applied site-specific recombination. Nucleic Acids Res. 24:4256-4262.
Chan, K.M., Y.T. Liu, C.H. Ma, M. Jayaram, and S. Sau. 2013. The 2 micron plasmid of Saccharomyces cerevisiae: a miniaturized selfish genome with optimized functional competence. Plasmid. 70:2-17.
Chen, Y., U. Narendra, L.E. Iype, M.M. Cox, and P.A. Rice. 2000. Crystal structure of a Flp recombinase-Holliday junction complex: assembly of an active oligomer by helix swapping. Mol Cell. 6:885-897.
Conway, A.B., Y. Chen, and P.A. Rice. 2003. Structural plasticity of the Flp-Holliday junction complex. J Mol Biol. 326:425-434.
Dymecki, S.M. 1996. Flp recombinase promotes site-specific DNA recombination in embryonic stem cells and transgenic mice. Proc Natl Acad Sci U S A. 93:6191-6196.
Gates, C.A., and M.M. Cox. 1988. FLP recombinase is an enzyme. Proc Natl Acad Sci U S A. 85:4628-4632.
Jahnz, M., and P. Schwille. 2005. An ultrasensitive site-specific DNA recombination assay based on dual-color fluorescence cross-correlation spectroscopy. Nucleic Acids Res. 33.
Jensen, I.S., K. Inui, S. Drakulic, S. Jayaprakash, B. Sander, and M.M. Golas. 2017. Expression of Flp Protein in a Baculovirus/Insect Cell System for Biotechnological Applications. Protein J. 36:332-342.
Kalderon, D., B.L. Roberts, W.D. Richardson, and A.E. Smith. 1984. A short amino acid sequence able to specify nuclear location. Cell. 39:499-509.
Kanki, H., H. Suzuki, and S. Itohara. 2006. High-efficiency CAG-FLPe deleter mice in C57BL/6J background. Exp Anim. 55:137-141.
Kranz, A., J. Fu, K. Duerschke, S. Weidlich, R. Naumann, A.F. Stewart, and K. Anastassiadis. 2010. An improved Flp deleter mouse in C57Bl/6 based on Flpo recombinase. Genesis. 48:512-520.
Laemmli, U.K. 1970. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227:680-685.
Low, B.E., P.M. Kutny, and M.V. Wiles. 2016. Simple, Efficient CRISPR-Cas9-Mediated Gene Editing in Mice: Strategies and Methods. Methods Mol Biol:3661-3668_3662.
Lu, C., and H.P. Erickson. 1997. Expression in Escherichia coli of the thermostable DNA polymerase from Pyrococcus furiosus. Protein Expr Purif. 11:179-184.
Ma, C.-H., B.-Y. Su, A. Maciaszek, H.-F. Fan, P. Guga, and M. Jayaram. 2019. A Flp-SUMO hybrid recombinase reveals multi-layered copy number control of a selfish DNA element through post-translational modification. PLOS Genetics. 15:e1008193.
Metje-Sprink, J., J. Menz, D. Modrzejewski, and T. Sprink. 2019. DNA-Free Genome Editing: Past, Present and Future. Front Plant Sci. 9.
Meyer-Leon, L., C.A. Gates, J.M. Attwood, E.A. Wood, and M.M. Cox. 1987. Purification of the FLP site-specific recombinase by affinity chromatography and re-examination of basic properties of the system. Nucleic Acids Res. 15:6469-6488.
Pan, H., D. Clary, and P.D. Sadowski. 1991. Identification of the DNA-binding domain of the FLP recombinase. J Biol Chem. 266:11347-11354.
Patsch, C., M. Peitz, D.M. Otte, D. Kesseler, J. Jungverdorben, F.T. Wunderlich, O. Brustle, A. Zimmer, and F. Edenhofer. 2010. Engineering cell-permeant FLP recombinase for tightly controlled inducible and reversible overexpression in embryonic stem cells. Stem Cells. 28:894-902.
Ponsaerts, P., J.P. Brown, D. Van den Plas, L. Van den Eeden, D.R. Van Bockstaele, P.G. Jorens, V.F. Van Tendeloo, J. Merregaert, P.B. Singh, and Z.N. Berneman. 2004. Messenger RNA electroporation is highly efficient in mouse embryonic stem cells: successful FLPe- and Cre-mediated recombination. Gene Ther. 11:1606-1610.
Raines, R.T., M. McCormick, T.R. Van Oosbree, and R.C. Mierendorf. 2000. The S.Tag fusion system for protein purification. Methods Enzymol. 326:362-376.
Raymond, C.S., and P. Soriano. 2007. High-efficiency FLP and PhiC31 site-specific recombination in mammalian cells. PLoS One. 2:0000162.
Raymond, C.S., and P. Soriano. 2010. ROSA26Flpo deleter mice promote efficient inversion of conditional gene traps in vivo. Genesis. 48:603-606.
Rodríguez, C.I., F. Buchholz, J. Galloway, R. Sequerra, J. Kasper, R. Ayala, A.F. Stewart, and S.M. Dymecki. High-efficiency deleter mice show that FLPe is an alternative to Cre-loxP. Nat Genet. 2000 Jun;25(2):139-40. doi: 10.1038/75973.
Schlake, T., and J. Bode. 1994. Use of mutated FLP recognition target (FRT) sites for the exchange of expression cassettes at defined chromosomal loci. Biochemistry. 33:12746-12751.
Singh, P., J.C. Schimenti, and E. Bolcun-Filas. 2015. A mouse geneticist's practical guide to CRISPR applications. Genetics. 199:1-15.
Titus, D.E. 1991. Cloning of DNA inserts. In Promega Protocols and Applications Guide. D.E. Titus, editor, USA. 52-53.
Turan, S., M. Galla, E. Ernst, J. Qiao, C. Voelkel, B. Schiedlmeier, C. Zehe, and J. Bode. 2011. Recombinase-mediated cassette exchange (RMCE): traditional concepts and current challenges. J Mol Biol. 407:193-221.
Turan, S., C. Zehe, J. Kuehle, J. Qiao, and J. Bode. 2013. Recombinase-mediated cassette exchange (RMCE) - a rapidly-expanding toolbox for targeted genomic modifications. Gene. 515:1-27.
Wu, Y., C. Wang, H. Sun, D. LeRoith, and S. Yakar. 2009. High-efficient FLPo deleter mice in C57BL/6J background. PLoS One. 4:0008054.
Xue, M., B.V. Atallah, and M. Scanziani. 2014. Equalizing excitation-inhibition ratios across visual cortical neurons. Nature. 511:596-600.
--->
<110> Федеральное государственное бюджетное учреждение науки Институт
биологии гена Российской академии наук (Institute of Gene Biology Russian
Academy of Sciences)
<120> Штамм клеток Escherichia coli BL21(DE3)/pET32v11-Flpo, продуцирующих
сайт-специфическую рекомбиназу Flpe
<160> 4
<210> 1
<211> 5596
<212> ДНК
<213> artificial sequence
<220>
<223> Плазмидная конструкция pET32v11
<400> 1
gatcccgcga aattaatacg actcactata ggggaattgt gagcggataa caattcccct 60
ctagaaataa ttttgtttaa ctttaagaag gagatataca tatgcaccat catcatcatc 120
attcttctgg tctggtgcca cgcggttctg gtatgaaaga aaccgctgct gctaaattcg 180
aacgccagca catggacagc ccagatctgg gtaccgacga cgacgacaag gccatgggca 240
gatctgaaaa cctgtacttt cagagcggat ccccggaatt cccgggtcga ctcgatcgag 300
cggccgcatc gtgactgact gacgatctcg agcaccacca ccaccaccac tgagatccgg 360
ctgctaacaa agcccgaaag gaagctgagt tggctgctgc caccgctgag caataactag 420
cataacccct tggggcctct aaacgggtct tgaggggttt tttgctgaaa ggaggaacta 480
tatccggatt ggcgaatggg acgcgccctg tagcggcgca ttaagcgcgg cgggtgtggt 540
ggttacgcgc agcgtgaccg ctacacttgc cagcgcccta gcgcccgctc ctttcgcttt 600
cttcccttcc tttctcgcca cgttcgccgg ctttccccgt caagctctaa atcgggggct 660
ccctttaggg ttccgattta gtgctttacg gcacctcgac cccaaaaaac ttgattaggg 720
tgatggttca cgtagtgggc catcgccctg atagacggtt tttcgccctt tgacgttgga 780
gtccacgttc tttaatagtg gactcttgtt ccaaactgga acaacactca accctatctc 840
ggtctattct tttgatttat aagggatttt gccgatttcg gcctattggt taaaaaatga 900
gctgatttaa caaaaattta acgcgaattt taacaaaata ttaacgttta caatttcagg 960
tggcactttt cggggaaatg tgcgcggaac ccctatttgt ttatttttct aaatacattc 1020
aaatatgtat ccgctcatga gacaataacc ctgataaatg cttcaataat attgaaaaag 1080
gaagagtatg agtattcaac atttccgtgt cgcccttatt cccttttttg cggcattttg 1140
ccttcctgtt tttgctcacc cagaaacgct ggtgaaagta aaagatgctg aagatcagtt 1200
gggtgcacga gtgggttaca tcgaactgga tctcaacagc ggtaagatcc ttgagagttt 1260
tcgccccgaa gaacgttttc caatgatgag cacttttaaa gttctgctat gtggcgcggt 1320
attatcccgt attgacgccg ggcaagagca actcggtcgc cgcatacact attctcagaa 1380
tgacttggtt gagtactcac cagtcacaga aaagcatctt acggatggca tgacagtaag 1440
agaattatgc agtgctgcca taaccatgag tgataacact gcggccaact tacttctgac 1500
aacgatcgga ggaccgaagg agctaaccgc ttttttgcac aacatggggg atcatgtaac 1560
tcgccttgat cgttgggaac cggagctgaa tgaagccata ccaaacgacg agcgtgacac 1620
cacgatgcct gcagcaatgg caacaacgtt gcgcaaacta ttaactggcg aactacttac 1680
tctagcttcc cggcaacaat taatagactg gatggaggcg gataaagttg caggaccact 1740
tctgcgctcg gcccttccgg ctggctggtt tattgctgat aaatctggag ccggtgagcg 1800
tgggtctcgc ggtatcattg cagcactggg gccagatggt aagccctccc gtatcgtagt 1860
tatctacacg acggggagtc aggcaactat ggatgaacga aatagacaga tcgctgagat 1920
aggtgcctca ctgattaagc attggtaact gtcagaccaa gtttactcat atatacttta 1980
gattgattta aaacttcatt tttaatttaa aaggatctag gtgaagatcc tttttgataa 2040
tctcatgacc aaaatccctt aacgtgagtt ttcgttccac tgagcgtcag accccgtaga 2100
aaagatcaaa ggatcttctt gagatccttt ttttctgcgc gtaatctgct gcttgcaaac 2160
aaaaaaacca ccgctaccag cggtggtttg tttgccggat caagagctac caactctttt 2220
tccgaaggta actggcttca gcagagcgca gataccaaat actgtccttc tagtgtagcc 2280
gtagttaggc caccacttca agaactctgt agcaccgcct acatacctcg ctctgctaat 2340
cctgttacca gtggctgctg ccagtggcga taagtcgtgt cttaccgggt tggactcaag 2400
acgatagtta ccggataagg cgcagcggtc gggctgaacg gggggttcgt gcacacagcc 2460
cagcttggag cgaacgacct acaccgaact gagataccta cagcgtgagc tatgagaaag 2520
cgccacgctt cccgaaggga gaaaggcgga caggtatccg gtaagcggca gggtcggaac 2580
aggagagcgc acgagggagc ttccaggggg aaacgcctgg tatctttata gtcctgtcgg 2640
gtttcgccac ctctgacttg agcgtcgatt tttgtgatgc tcgtcagggg ggcggagcct 2700
atggaaaaac gccagcaacg cggccttttt acggttcctg gccttttgct ggccttttgc 2760
tcacatgttc tttcctgcgt tatcccctga ttctgtggat aaccgtatta ccgcctttga 2820
gtgagctgat accgctcgcc gcagccgaac gaccgagcgc agcgagtcag tgagcgagga 2880
agcggaagag cgcctgatgc ggtattttct ccttacgcat ctgtgcggta tttcacaccg 2940
catatatggt gcactctcag tacaatctgc tctgatgccg catagttaag ccagtataca 3000
ctccgctatc gctacgtgac tgggtcatgg ctgcgccccg acacccgcca acacccgctg 3060
acgcgccctg acgggcttgt ctgctcccgg catccgctta cagacaagct gtgaccgtct 3120
ccgggagctg catgtgtcag aggttttcac cgtcatcacc gaaacgcgcg aggcagctgc 3180
ggtaaagctc atcagcgtgg tcgtgaagcg attcacagat gtctgcctgt tcatccgcgt 3240
ccagctcgtt gagtttctcc agaagcgtta atgtctggct tctgataaag cgggccatgt 3300
taagggcggt tttttcctgt ttggtcactg atgcctccgt gtaaggggga tttctgttca 3360
tgggggtaat gataccgatg aaacgagaga ggatgctcac gatacgggtt actgatgatg 3420
aacatgcccg gttactggaa cgttgtgagg gtaaacaact ggcggtatgg atgcggcggg 3480
accagagaaa aatcactcag ggtcaatgcc agcgcttcgt taatacagat gtaggtgttc 3540
cacagggtag ccagcagcat cctgcgatgc agatccggaa cataatggtg cagggcgctg 3600
acttccgcgt ttccagactt tacgaaacac ggaaaccgaa gaccattcat gttgttgctc 3660
aggtcgcaga cgttttgcag cagcagtcgc ttcacgttcg ctcgcgtatc ggtgattcat 3720
tctgctaacc agtaaggcaa ccccgccagc ctagccgggt cctcaacgac aggagcacga 3780
tcatgcgcac ccgtggggcc gccatgccgg cgataatggc ctgcttctcg ccgaaacgtt 3840
tggtggcggg accagtgacg aaggcttgag cgagggcgtg caagattccg aataccgcaa 3900
gcgacaggcc gatcatcgtc gcgctccagc gaaagcggtc ctcgccgaaa atgacccaga 3960
gcgctgccgg cacctgtcct acgagttgca tgataaagaa gacagtcata agtgcggcga 4020
cgatagtcat gccccgcgcc caccggaagg agctgactgg gttgaaggct ctcaagggca 4080
tcggtcgaga tcccggtgcc taatgagtga gctaacttac attaattgcg ttgcgctcac 4140
tgcccgcttt ccagtcggga aacctgtcgt gccagctgca ttaatgaatc ggccaacgcg 4200
cggggagagg cggtttgcgt attgggcgcc agggtggttt ttcttttcac cagtgagacg 4260
ggcaacagct gattgccctt caccgcctgg ccctgagaga gttgcagcaa gcggtccacg 4320
ctggtttgcc ccagcaggcg aaaatcctgt ttgatggtgg ttaacggcgg gatataacat 4380
gagctgtctt cggtatcgtc gtatcccact accgagatgt ccgcaccaac gcgcagcccg 4440
gactcggtaa tggcgcgcat tgcgcccagc gccatctgat cgttggcaac cagcatcgca 4500
gtgggaacga tgccctcatt cagcatttgc atggtttgtt gaaaaccgga catggcactc 4560
cagtcgcctt cccgttccgc tatcggctga atttgattgc gagtgagata tttatgccag 4620
ccagccagac gcagacgcgc cgagacagaa cttaatgggc ccgctaacag cgcgatttgc 4680
tggtgaccca atgcgaccag atgctccacg cccagtcgcg taccgtcttc atgggagaaa 4740
ataatactgt tgatgggtgt ctggtcagag acatcaagaa ataacgccgg aacattagtg 4800
caggcagctt ccacagcaat ggcatcctgg tcatccagcg gatagttaat gatcagccca 4860
ctgacgcgtt gcgcgagaag attgtgcacc gccgctttac aggcttcgac gccgcttcgt 4920
tctaccatcg acaccaccac gctggcaccc agttgatcgg cgcgagattt aatcgccgcg 4980
acaatttgcg acggcgcgtg cagggccaga ctggaggtgg caacgccaat cagcaacgac 5040
tgtttgcccg ccagttgttg tgccacgcgg ttgggaatgt aattcagctc cgccatcgcc 5100
gcttccactt tttcccgcgt tttcgcagaa acgtggctgg cctggttcac cacgcgggaa 5160
acggtctgat aagagacacc ggcatactct gcgacatcgt ataacgttac tggtttcaca 5220
ttcaccaccc tgaattgact ctcttccggg cgctatcatg ccataccgcg aaaggttttg 5280
cgccattcga tggtgtccgg gatctcgacg ctctccctta tgcgactcct gcattaggaa 5340
gcagcccagt agtaggttga ggccgttgag caccgccgcc gcaaggaatg gtgcatgcaa 5400
ggagatggcg cccaacagtc ccccggccac ggggcctgcc accataccca cgccgaaaca 5460
agcgctcatg agcccgaagt ggcgagcccg atcttcccca tcggtgatgt cggcgatata 5520
ggcgccagca accgcacctg tggcgccggt gatgccggcc acgatgcgtc cggcgtagag 5580
gatcgagatc gatctc 5596
<210> 2
<211> 6888
<212> ДНК
<213> artificial sequence
<220>
<223> Плазмидный вектор pET32v11-Flpo
<400> 2
gatcccgcga aattaatacg actcactata ggggaattgt gagcggataa caattcccct 60
ctagaaataa ttttgtttaa ctttaagaag gagatataca tatgcaccat catcatcatc 120
attcttctgg tctggtgcca cgcggttctg gtatgaaaga aaccgctgct gctaaattcg 180
aacgccagca catggacagc ccagatctgg gtaccgacga cgacgacaag gccatgggca 240
gatctgaaaa cctgtacttt cagagcggat ccccggaatt cgctcctaag aagaagagga 300
aggtgatgag ccagttcgac atcctgtgca agaccccccc caaggtgctg gtgcggcagt 360
tcgtggagag attcgagagg cccagcggcg agaagatcgc cagctgtgcc gccgagctga 420
cctacctgtg ctggatgatc acccacaacg gcaccgccat caagagggcc accttcatga 480
gctacaacac catcatcagc aacagcctga gcttcgacat cgtgaacaag agcctgcagt 540
tcaagtacaa gacccagaag gccaccatcc tggaggccag cctgaagaag ctgatccccg 600
cctgggagtt caccatcatc ccttacaacg gccagaagca ccagagcgac atcaccgaca 660
tcgtgtccag cctgcagctg cagttcgaga gcagcgagga ggccgacaag ggcaacagcc 720
acagcaagaa gatgctgaag gccctgctgt ccgagggcga gagcatctgg gagatcaccg 780
agaagatcct gaacagcttc gagtacacca gcaggttcac caagaccaag accctgtacc 840
agttcctgtt cctggccaca ttcatcaact gcggcaggtt cagcgacatc aagaacgtgg 900
accccaagag cttcaagctg gtgcagaaca agtacctggg cgtgatcatt cagtgcctgg 960
tgaccgagac caagacaagc gtgtccaggc acatctactt tttcagcgcc agaggcagga 1020
tcgaccccct ggtgtacctg gacgagttcc tgaggaacag cgagcccgtg ctgaagagag 1080
tgaacaggac cggcaacagc agcagcaaca agcaggagta ccagctgctg aaggacaacc 1140
tggtgcgcag ctacaacaag gccctgaaga agaacgcccc ctaccccatc ttcgctatca 1200
agaacggccc taagagccac atcggcaggc acctgatgac cagctttctg agcatgaagg 1260
gcctgaccga gctgacaaac gtggtgggca actggagcga caagagggcc tccgccgtgg 1320
ccaggaccac ctacacccac cagatcaccg ccatccccga ccactacttc gccctggtgt 1380
ccaggtacta cgcctacgac cccatcagca aggagatgat cgccctgaag gacgagacca 1440
accccatcga ggagtggcag cacatcgagc agctgaaggg cagcgccgag ggcagcatca 1500
gataccccgc ctggaacggc atcatcagcc aggaggtgct ggactacctg agcagctaca 1560
tcaacaggcg gatctgagtc gactcgatcg agcggccgca tcgtgactga ctgacgatct 1620
cgagcaccac caccaccacc actgagatcc ggctgctaac aaagcccgaa aggaagctga 1680
gttggctgct gccaccgctg agcaataact agcataaccc cttggggcct ctaaacgggt 1740
cttgaggggt tttttgctga aaggaggaac tatatccgga ttggcgaatg ggacgcgccc 1800
tgtagcggcg cattaagcgc ggcgggtgtg gtggttacgc gcagcgtgac cgctacactt 1860
gccagcgccc tagcgcccgc tcctttcgct ttcttccctt cctttctcgc cacgttcgcc 1920
ggctttcccc gtcaagctct aaatcggggg ctccctttag ggttccgatt tagtgcttta 1980
cggcacctcg accccaaaaa acttgattag ggtgatggtt cacgtagtgg gccatcgccc 2040
tgatagacgg tttttcgccc tttgacgttg gagtccacgt tctttaatag tggactcttg 2100
ttccaaactg gaacaacact caaccctatc tcggtctatt cttttgattt ataagggatt 2160
ttgccgattt cggcctattg gttaaaaaat gagctgattt aacaaaaatt taacgcgaat 2220
tttaacaaaa tattaacgtt tacaatttca ggtggcactt ttcggggaaa tgtgcgcgga 2280
acccctattt gtttattttt ctaaatacat tcaaatatgt atccgctcat gagacaataa 2340
ccctgataaa tgcttcaata atattgaaaa aggaagagta tgagtattca acatttccgt 2400
gtcgccctta ttcccttttt tgcggcattt tgccttcctg tttttgctca cccagaaacg 2460
ctggtgaaag taaaagatgc tgaagatcag ttgggtgcac gagtgggtta catcgaactg 2520
gatctcaaca gcggtaagat ccttgagagt tttcgccccg aagaacgttt tccaatgatg 2580
agcactttta aagttctgct atgtggcgcg gtattatccc gtattgacgc cgggcaagag 2640
caactcggtc gccgcataca ctattctcag aatgacttgg ttgagtactc accagtcaca 2700
gaaaagcatc ttacggatgg catgacagta agagaattat gcagtgctgc cataaccatg 2760
agtgataaca ctgcggccaa cttacttctg acaacgatcg gaggaccgaa ggagctaacc 2820
gcttttttgc acaacatggg ggatcatgta actcgccttg atcgttggga accggagctg 2880
aatgaagcca taccaaacga cgagcgtgac accacgatgc ctgcagcaat ggcaacaacg 2940
ttgcgcaaac tattaactgg cgaactactt actctagctt cccggcaaca attaatagac 3000
tggatggagg cggataaagt tgcaggacca cttctgcgct cggcccttcc ggctggctgg 3060
tttattgctg ataaatctgg agccggtgag cgtgggtctc gcggtatcat tgcagcactg 3120
gggccagatg gtaagccctc ccgtatcgta gttatctaca cgacggggag tcaggcaact 3180
atggatgaac gaaatagaca gatcgctgag ataggtgcct cactgattaa gcattggtaa 3240
ctgtcagacc aagtttactc atatatactt tagattgatt taaaacttca tttttaattt 3300
aaaaggatct aggtgaagat cctttttgat aatctcatga ccaaaatccc ttaacgtgag 3360
ttttcgttcc actgagcgtc agaccccgta gaaaagatca aaggatcttc ttgagatcct 3420
ttttttctgc gcgtaatctg ctgcttgcaa acaaaaaaac caccgctacc agcggtggtt 3480
tgtttgccgg atcaagagct accaactctt tttccgaagg taactggctt cagcagagcg 3540
cagataccaa atactgtcct tctagtgtag ccgtagttag gccaccactt caagaactct 3600
gtagcaccgc ctacatacct cgctctgcta atcctgttac cagtggctgc tgccagtggc 3660
gataagtcgt gtcttaccgg gttggactca agacgatagt taccggataa ggcgcagcgg 3720
tcgggctgaa cggggggttc gtgcacacag cccagcttgg agcgaacgac ctacaccgaa 3780
ctgagatacc tacagcgtga gctatgagaa agcgccacgc ttcccgaagg gagaaaggcg 3840
gacaggtatc cggtaagcgg cagggtcgga acaggagagc gcacgaggga gcttccaggg 3900
ggaaacgcct ggtatcttta tagtcctgtc gggtttcgcc acctctgact tgagcgtcga 3960
tttttgtgat gctcgtcagg ggggcggagc ctatggaaaa acgccagcaa cgcggccttt 4020
ttacggttcc tggccttttg ctggcctttt gctcacatgt tctttcctgc gttatcccct 4080
gattctgtgg ataaccgtat taccgccttt gagtgagctg ataccgctcg ccgcagccga 4140
acgaccgagc gcagcgagtc agtgagcgag gaagcggaag agcgcctgat gcggtatttt 4200
ctccttacgc atctgtgcgg tatttcacac cgcatatatg gtgcactctc agtacaatct 4260
gctctgatgc cgcatagtta agccagtata cactccgcta tcgctacgtg actgggtcat 4320
ggctgcgccc cgacacccgc caacacccgc tgacgcgccc tgacgggctt gtctgctccc 4380
ggcatccgct tacagacaag ctgtgaccgt ctccgggagc tgcatgtgtc agaggttttc 4440
accgtcatca ccgaaacgcg cgaggcagct gcggtaaagc tcatcagcgt ggtcgtgaag 4500
cgattcacag atgtctgcct gttcatccgc gtccagctcg ttgagtttct ccagaagcgt 4560
taatgtctgg cttctgataa agcgggccat gttaagggcg gttttttcct gtttggtcac 4620
tgatgcctcc gtgtaagggg gatttctgtt catgggggta atgataccga tgaaacgaga 4680
gaggatgctc acgatacggg ttactgatga tgaacatgcc cggttactgg aacgttgtga 4740
gggtaaacaa ctggcggtat ggatgcggcg ggaccagaga aaaatcactc agggtcaatg 4800
ccagcgcttc gttaatacag atgtaggtgt tccacagggt agccagcagc atcctgcgat 4860
gcagatccgg aacataatgg tgcagggcgc tgacttccgc gtttccagac tttacgaaac 4920
acggaaaccg aagaccattc atgttgttgc tcaggtcgca gacgttttgc agcagcagtc 4980
gcttcacgtt cgctcgcgta tcggtgattc attctgctaa ccagtaaggc aaccccgcca 5040
gcctagccgg gtcctcaacg acaggagcac gatcatgcgc acccgtgggg ccgccatgcc 5100
ggcgataatg gcctgcttct cgccgaaacg tttggtggcg ggaccagtga cgaaggcttg 5160
agcgagggcg tgcaagattc cgaataccgc aagcgacagg ccgatcatcg tcgcgctcca 5220
gcgaaagcgg tcctcgccga aaatgaccca gagcgctgcc ggcacctgtc ctacgagttg 5280
catgataaag aagacagtca taagtgcggc gacgatagtc atgccccgcg cccaccggaa 5340
ggagctgact gggttgaagg ctctcaaggg catcggtcga gatcccggtg cctaatgagt 5400
gagctaactt acattaattg cgttgcgctc actgcccgct ttccagtcgg gaaacctgtc 5460
gtgccagctg cattaatgaa tcggccaacg cgcggggaga ggcggtttgc gtattgggcg 5520
ccagggtggt ttttcttttc accagtgaga cgggcaacag ctgattgccc ttcaccgcct 5580
ggccctgaga gagttgcagc aagcggtcca cgctggtttg ccccagcagg cgaaaatcct 5640
gtttgatggt ggttaacggc gggatataac atgagctgtc ttcggtatcg tcgtatccca 5700
ctaccgagat gtccgcacca acgcgcagcc cggactcggt aatggcgcgc attgcgccca 5760
gcgccatctg atcgttggca accagcatcg cagtgggaac gatgccctca ttcagcattt 5820
gcatggtttg ttgaaaaccg gacatggcac tccagtcgcc ttcccgttcc gctatcggct 5880
gaatttgatt gcgagtgaga tatttatgcc agccagccag acgcagacgc gccgagacag 5940
aacttaatgg gcccgctaac agcgcgattt gctggtgacc caatgcgacc agatgctcca 6000
cgcccagtcg cgtaccgtct tcatgggaga aaataatact gttgatgggt gtctggtcag 6060
agacatcaag aaataacgcc ggaacattag tgcaggcagc ttccacagca atggcatcct 6120
ggtcatccag cggatagtta atgatcagcc cactgacgcg ttgcgcgaga agattgtgca 6180
ccgccgcttt acaggcttcg acgccgcttc gttctaccat cgacaccacc acgctggcac 6240
ccagttgatc ggcgcgagat ttaatcgccg cgacaatttg cgacggcgcg tgcagggcca 6300
gactggaggt ggcaacgcca atcagcaacg actgtttgcc cgccagttgt tgtgccacgc 6360
ggttgggaat gtaattcagc tccgccatcg ccgcttccac tttttcccgc gttttcgcag 6420
aaacgtggct ggcctggttc accacgcggg aaacggtctg ataagagaca ccggcatact 6480
ctgcgacatc gtataacgtt actggtttca cattcaccac cctgaattga ctctcttccg 6540
ggcgctatca tgccataccg cgaaaggttt tgcgccattc gatggtgtcc gggatctcga 6600
cgctctccct tatgcgactc ctgcattagg aagcagccca gtagtaggtt gaggccgttg 6660
agcaccgccg ccgcaaggaa tggtgcatgc aaggagatgg cgcccaacag tcccccggcc 6720
acggggcctg ccaccatacc cacgccgaaa caagcgctca tgagcccgaa gtggcgagcc 6780
cgatcttccc catcggtgat gtcggcgata taggcgccag caaccgcacc tgtggcgccg 6840
gtgatgccgg ccacgatgcg tccggcgtag aggatcgaga tcgatctc 6888
<210> 3
<211> 491
<212> PRT
<213> artificial sequence
<220>
<223> 6-His-S-Flpe
<400> 3
Met His His His His His His Ser Ser Gly Leu Val Pro Arg Gly Ser 16
5 10 15
Gly Met Lys Glu Thr Ala Ala Ala Lys Phe Glu Arg Gln His Met Asp 32
20 25 30
Ser Pro Asp Leu Gly Thr Asp Asp Asp Asp Lys Ala Met Gly Arg Ser 48
35 40 45
Glu Asn Leu Tyr Phe Gln Ser Gly Ser Pro Glu Phe Ala Pro Lys Lys 64
50 55 60
Lys Arg Lys Val Met Ser Gln Phe Asp Ile Leu Cys Lys Thr Pro Pro 80
65 70 75 80
Lys Val Leu Val Arg Gln Phe Val Glu Arg Phe Glu Arg Pro Ser Gly 96
85 90 95
Glu Lys Ile Ala Ser Cys Ala Ala Glu Leu Thr Tyr Leu Cys Trp Met 112
100 105 110
Ile Thr His Asn Gly Thr Ala Ile Lys Arg Ala Thr Phe Met Ser Tyr 128
115 120 125
Asn Thr Ile Ile Ser Asn Ser Leu Ser Phe Asp Ile Val Asn Lys Ser 144
130 135 140
Leu Gln Phe Lys Tyr Lys Thr Gln Lys Ala Thr Ile Leu Glu Ala Ser 160
145 150 155 160
Leu Lys Lys Leu Ile Pro Ala Trp Glu Phe Thr Ile Ile Pro Tyr Asn 176
165 170 175
Gly Gln Lys His Gln Ser Asp Ile Thr Asp Ile Val Ser Ser Leu Gln 192
180 185 190
Leu Gln Phe Glu Ser Ser Glu Glu Ala Asp Lys Gly Asn Ser His Ser 208
195 200 205
Lys Lys Met Leu Lys Ala Leu Leu Ser Glu Gly Glu Ser Ile Trp Glu 224
210 215 220
Ile Thr Glu Lys Ile Leu Asn Ser Phe Glu Tyr Thr Ser Arg Phe Thr 240
225 230 235 240
Lys Thr Lys Thr Leu Tyr Gln Phe Leu Phe Leu Ala Thr Phe Ile Asn 256
245 250 255
Cys Gly Arg Phe Ser Asp Ile Lys Asn Val Asp Pro Lys Ser Phe Lys 272
260 265 270
Leu Val Gln Asn Lys Tyr Leu Gly Val Ile Ile Gln Cys Leu Val Thr 288
275 280 285
Glu Thr Lys Thr Ser Val Ser Arg His Ile Tyr Phe Phe Ser Ala Arg 304
290 295 300
Gly Arg Ile Asp Pro Leu Val Tyr Leu Asp Glu Phe Leu Arg Asn Ser 320
305 310 315 320
Glu Pro Val Leu Lys Arg Val Asn Arg Thr Gly Asn Ser Ser Ser Asn 336
325 330 335
Lys Gln Glu Tyr Gln Leu Leu Lys Asp Asn Leu Val Arg Ser Tyr Asn 352
340 345 350
Lys Ala Leu Lys Lys Asn Ala Pro Tyr Pro Ile Phe Ala Ile Lys Asn 368
355 360 365
Gly Pro Lys Ser His Ile Gly Arg His Leu Met Thr Ser Phe Leu Ser 384
370 375 380
Met Lys Gly Leu Thr Glu Leu Thr Asn Val Val Gly Asn Trp Ser Asp 400
385 390 395 400
Lys Arg Ala Ser Ala Val Ala Arg Thr Thr Tyr Thr His Gln Ile Thr 416
405 410 415
Ala Ile Pro Asp His Tyr Phe Ala Leu Val Ser Arg Tyr Tyr Ala Tyr 432
420 425 430
Asp Pro Ile Ser Lys Glu Met Ile Ala Leu Lys Asp Glu Thr Asn Pro 448
435 440 445
Ile Glu Glu Trp Gln His Ile Glu Gln Leu Lys Gly Ser Ala Glu Gly 464
450 455 460
Ser Ile Arg Tyr Pro Ala Trp Asn Gly Ile Ile Ser Gln Glu Val Leu 480
465 470 475 480
Asp Tyr Leu Ser Ser Tyr Ile Asn Arg Arg Ile 491
485 490
<210> 4
<211> 4287
<212> ДНК
<213> artificial sequence
<220>
<223> Плазмидная конструкция pSK-DsRed-2xFRT
<400> 4
gcaccccagg ctttacactt tatgcttccg gctcgtatgt tgtgtggaat tgtgagcgga 60
taacaatttc acacaggaaa cagctatgac catgattacg ccaagcgcgc aattaaccct 120
cactaaaggg aacaaaagct ggagctccac cgcggtggcg gccgctctag aactagtgga 180
tcccccggaa ttccttcgcc agggggatct tgaagttcct attccgaagt tcctattctt 240
caaatagtat aggaacttca agcttatcgg ggctgcagga attcgatgaa gttatcgtac 300
gggatctaat tcaattagag actaattcaa ttagagctaa ttcaattagg atccaagctt 360
atcgatttcg aaccctcgac cgccggagta taaatagagg cgcttcgtct acggagcgac 420
aattcaattc aaacaagcaa agtgaacacg tcgctaagcg aaagctaagc aaataaacaa 480
gcgcagctga acaagctaaa caatcggctc gaagccggtc gccaccatgg cctcctccga 540
ggacgtcatc aaggagttca tgcgcttcaa ggtgcgcatg gagggctccg tgaacggcca 600
cgagttcgag atcgagggcg agggcgaggg ccgcccctac gagggcaccc agaccgccaa 660
gctgaaggtg accaagggcg gccccctgcc cttcgcctgg gacatcctgt ccccccagtt 720
ccagtacggc tccaaggtgt acgtgaagca ccccgccgac atccccgact acaagaagct 780
gtccttcccc gagggcttca agtgggagcg cgtgatgaac ttcgaggacg gcggcgtggt 840
gaccgtgacc caggactcct ccctccagga cggctccttc atctacaagg tgaagttcat 900
cggcgtgaac ttcccctccg acggccccgt aatgcagaag aagactatgg gctgggaggc 960
gtccaccgag cgcctgtacc cccgcgacgg cgtgctgaag ggcgagatcc acaaggccct 1020
gaagctgaag gacggcggcc actacctggt ggagttcaag tccatctaca tggccaagaa 1080
gcccgtgcag ctgcccggct actactacgt ggactccaag ctggacatca cctcccacaa 1140
cgaggactac accatcgtgg agcagtacga gcgcgccgag ggccgccacc acctgttcct 1200
gtaggggccg cgactctaga tcataatcag ccataccaca tttgtagagg ttttacttgc 1260
tttaaaaaac ctcccacacc tccccctgaa cctgaaacat aaaatgaatg caattgttgt 1320
tgttaacttg tttattgcag cttataatgg ttacaaataa agcaatagca tcacaaattt 1380
cacaaataaa gcattttttt cactgcattc tagttgtggt ttgtccaaac tcatcaatgt 1440
atcatcaagc ttatcgatac cgtcgaggaa ttccttcgcc agggggatct tgaagttcct 1500
attccgaagt tcctattctt caaatagtat aggaacttca agcttatcgt cgacctcgag 1560
ggggggcccg gtacccaatt cgccctatag tgagtcgtat tacgcgcgct cactggccgt 1620
cgttttacaa cgtcgtgact gggaaaaccc tggcgttacc caacttaatc gccttgcagc 1680
acatccccct ttcgccagct ggcgtaatag cgaagaggcc cgcaccgatc gcccttccca 1740
acagttgcgc agcctgaatg gcgaatggga cgcgccctgt agcggcgcat taagcgcggc 1800
gggtgtggtg gttacgcgca gcgtgaccgc tacacttgcc agcgccctag cgcccgctcc 1860
tttcgctttc ttcccttcct ttctcgccac gttcgccggc tttccccgtc aagctctaaa 1920
tcgggggctc cctttagggt tccgatttag tgctttacgg cacctcgacc ccaaaaaact 1980
tgattagggt gatggttcac gtagtgggcc atcgccctga tagacggttt ttcgcccttt 2040
gacgttggag tccacgttct ttaatagtgg actcttgttc caaactggaa caacactcaa 2100
ccctatctcg gtctattctt ttgatttata agggattttg ccgatttcgg cctattggtt 2160
aaaaaatgag ctgatttaac aaaaatttaa cgcgaatttt aacaaaatat taacgcttac 2220
aatttaggtg gcacttttcg gggaaatgtg cgcggaaccc ctatttgttt atttttctaa 2280
atacattcaa atatgtatcc gctcatgaga caataaccct gataaatgct tcaataatat 2340
tgaaaaagga agagtatgag tattcaacat ttccgtgtcg cccttattcc cttttttgcg 2400
gcattttgcc ttcctgtttt tgctcaccca gaaacgctgg tgaaagtaaa agatgctgaa 2460
gatcagttgg gtgcacgagt gggttacatc gaactggatc tcaacagcgg taagatcctt 2520
gagagttttc gccccgaaga acgttttcca atgatgagca cttttaaagt tctgctatgt 2580
ggcgcggtat tatcccgtat tgacgccggg caagagcaac tcggtcgccg catacactat 2640
tctcagaatg acttggttga gtactcacca gtcacagaaa agcatcttac ggatggcatg 2700
acagtaagag aattatgcag tgctgccata accatgagtg ataacactgc ggccaactta 2760
cttctgacaa cgatcggagg accgaaggag ctaaccgctt ttttgcacaa catgggggat 2820
catgtaactc gccttgatcg ttgggaaccg gagctgaatg aagccatacc aaacgacgag 2880
cgtgacacca cgatgcctgt agcaatggca acaacgttgc gcaaactatt aactggcgaa 2940
ctacttactc tagcttcccg gcaacaatta atagactgga tggaggcgga taaagttgca 3000
ggaccacttc tgcgctcggc ccttccggct ggctggttta ttgctgataa atctggagcc 3060
ggtgagcgtg ggtctcgcgg tatcattgca gcactggggc cagatggtaa gccctcccgt 3120
atcgtagtta tctacacgac ggggagtcag gcaactatgg atgaacgaaa tagacagatc 3180
gctgagatag gtgcctcact gattaagcat tggtaactgt cagaccaagt ttactcatat 3240
atactttaga ttgatttaaa acttcatttt taatttaaaa ggatctaggt gaagatcctt 3300
tttgataatc tcatgaccaa aatcccttaa cgtgagtttt cgttccactg agcgtcagac 3360
cccgtagaaa agatcaaagg atcttcttga gatccttttt ttctgcgcgt aatctgctgc 3420
ttgcaaacaa aaaaaccacc gctaccagcg gtggtttgtt tgccggatca agagctacca 3480
actctttttc cgaaggtaac tggcttcagc agagcgcaga taccaaatac tgtccttcta 3540
gtgtagccgt agttaggcca ccacttcaag aactctgtag caccgcctac atacctcgct 3600
ctgctaatcc tgttaccagt ggctgctgcc agtggcgata agtcgtgtct taccgggttg 3660
gactcaagac gatagttacc ggataaggcg cagcggtcgg gctgaacggg gggttcgtgc 3720
acacagccca gcttggagcg aacgacctac accgaactga gatacctaca gcgtgagcta 3780
tgagaaagcg ccacgcttcc cgaagggaga aaggcggaca ggtatccggt aagcggcagg 3840
gtcggaacag gagagcgcac gagggagctt ccagggggaa acgcctggta tctttatagt 3900
cctgtcgggt ttcgccacct ctgacttgag cgtcgatttt tgtgatgctc gtcagggggg 3960
cggagcctat ggaaaaacgc cagcaacgcg gcctttttac ggttcctggc cttttgctgg 4020
ccttttgctc acatgttctt tcctgcgtta tcccctgatt ctgtggataa ccgtattacc 4080
gcctttgagt gagctgatac cgctcgccgc agccgaacga ccgagcgcag cgagtcagtg 4140
agcgaggaag cggaagagcg cccaatacgc aaaccgcctc tccccgcgcg ttggccgatt 4200
cattaatgca gctggcacga caggtttccc gactggaaag cgggcagtga gcgcaacgca 4260
attaatgtga gttagctcac tcattag 4286
<---
Изобретение относится к области генной инженерии и молекулярной биологии. Предложена рекомбинантная плазмида pET32v11-Flpo, имеющая последовательность SEQ ID NO: 2, обеспечивающая синтез рекомбинантного белка 6His-S-Flpe последовательностью SEQ ID NO: 3 в клетках Escherichia coli и содержащая ген рекомбиназы Flpo, который включает в себя сайты расщепления для протеаз тромбина, энтерокиназы и TEV протеазы вируса табачной мозаики. При этом указанная плазмида получена путем клонирования кДНК рекомбиназы Flpo в вектор pET32v11, а кДНК Flpo с N-концевой последовательностью ядерной локализации NLS находится в одной рамке считывания с 6-гистидиновым и S-эпитопами и аминокислотными последовательностями сайтов расщепления тромбина, энтерокиназы и протеазы TEV. Также предложен штамм Escherichia coli BL21(DE3)/pET32v11-Flpo, продуцирующий рекомбинантный белок 6His-S-Flpe последовательностью SEQ ID NO: 3 и полученный путем трансформации клеток штамма Escherichia coli BL21(DE3) вышеуказанной плазмидой. Изобретение позволяет получить с высоким выходом каталитически активную рекомбиназу Flpe. 2 н.п. ф-лы, 6 ил., 5 пр.
1. Рекомбинантная плазмида pET32v11-Flpo, имеющая последовательность SEQ ID NO: 2, обеспечивающая синтез рекомбинантного белка 6His-S-Flpe последовательностью SEQ ID NO: 3 в клетках Escherichia coli и содержащая ген рекомбиназы Flpo, который включает в себя сайты расщепления для протеаз тромбина, энтерокиназы и TEV протеазы вируса табачной мозаики, которая получена путем клонирования кДНК рекомбиназы Flpo в вектор pET32v11, отличающаяся тем, что кДНК Flpo с N-концевой последовательностью ядерной локализации NLS находится в одной рамке считывания с 6-гистидиновым и S-эпитопами и аминокислотными последовательностями сайтов расщепления тромбина, энтерокиназы и протеазы TEV.
2. Штамм Escherichia coli BL21(DE3)/pET32v11-Flpo, продуцирующий рекомбинантный белок 6His-S-Flpe последовательностью SEQ ID NO: 3 и полученный путем трансформации клеток штамма Escherichia coli BL21(DE3) плазмидой по п. 1.
| CONWAY A.B | |||
| et al | |||
| Structural plasticity of the Flp-Holliday junction complex | |||
| J Mol Biol | |||
| Способ и приспособление для нагревания хлебопекарных камер | 1923 |
|
SU2003A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Найдено онлайн: https://www.sciencedirect.com/science/article/pii/S0022283602013700?via%3Dihub Дата обращения 21.07.2021 | |||
| JAHNZ M, SCHWILLE P | |||
| An ultrasensitive site-specific DNA recombination | |||
Авторы
Даты
2021-12-13—Публикация
2020-12-09—Подача