Изобретение относится к биотехнологии, в частности к технологии получения рекомбинантной нуклеазы Cpf1 с помощью штамма бактерий Escherichia coli.
Бактериальный белок Cpf1 является эффекторным белком системы CRISPR, который в комплексе с направляющей (guide) РНК (далее gPHK) генерирует двуцепочечный сайт-специфичный разрыв ДНК с образованием "липких" концов. Нуклеаза CRISPR-Cas Cpf1 (также известная как Casl2a нуклеаза) является аналогом нуклеазы Cas9 и обладает следующими отличительными свойствами: распознавание Т-богатого РАМ-сайта - последовательности, смежной с последовательностью мишени, наличие которой необходимо для нацеливания на мишень; эндонуклеазная активность, при которой в ДНК генерируются липкие 5'-концы ДНК (а не тупые двуцепочечное разрывы, как в случае Cas9); последовательность ДНК расщепляется на дистальном конце от РАМ-сайта (по сравнению с проксимальным расщеплением у Cas9), что уменьшает вероятность нарушения РАМ-сайта и дает возможность проведения нескольких генетических манипуляций в одном месте [1, 2].
Аналоги на получение штаммов-продуцентов эффекторных белков системы CRISPR/Cas, к которым относятся нуклеазы Cpf1 и Cas9, не выявлены. Ближайшими аналогами являются патенты на получение различных эндонуклеаз рестрикции (например, RU 2529362 C1, RU 2475533 C1). Эндонуклеазы рестрикции, так же как и эффекторные белки системы CRISPR/Cas, используются для генно-инженерных манипуляций и вносят в ДНК двуцепочечные разрывы, однако обладают рядом недостатков, таких как распознавание коротких 6-нуклеотидных последовательностей и неизменная специфичность (распознавание определенной единственной последовательности нуклеотидов). Предлагаемое изобретение решает данные проблемы, поскольку нуклеаза Cpf1 узнает 20-25-нуклеотидную последовательность, что важно при редактировании протяженных ДНК, так вероятность встретить несколько идентичных 6-нуклеотидных последовательностей выше примерно в 106-109 раз. Специфичность к мишени у Cpf1 задается через небольшую молекулу РНК, которую легко синтезировать химическим путем, поэтому один и тот же фермент может применяться для редактирования различных мишеней в отличие от эндонуклеаз рестрикции.
Задачей, решаемой авторами, являлось создание технологии высокоэффективной продукции активного рекомбинантного фермента Cpf1.
Технический результат был достигнут созданием штамма E. coli BL21(DE3)pLysS/pET15b-HisCpf1 - продуцента активной рекомбинантной нуклеазы Cpf1, полученного трансформацией культуры клеток E. coli BL21(DE3)pLysS (генотип F-, ompT, hsdSB (rB- mB-), dcm, gal, λ(DE3), pLysS, Cmr., рекомбинантной плазмидной ДНК pET15b-HisCpf1, сконструированной на основе вектора pET15b, несущего ген рекомбинантной нуклеазы Cpf1 из бактериального штамма Moraxella bovis.
Штамм Е. coli BL21(DE3)pLysS/pET15b-HisCpf1 имеет следующие характеристики:
Культурально-морфологические особенности штамма: грамотрицательные прямые палочки, размером 1,1-1,5×2,0-3,0 мкм, одиночные, спор и капсул не образуют. Каталазоположительные. Оксидазоотрицательные. Факультативные анаэробы.
Клетки хорошо растут на простых питательных средах, содержащих и не содержащих хлорамфеникол и ампициллин, например, на среде LB (питательная среда LisogenyBroth). На агаризованной среде - колонии гладкие, круглые, слабо выпуклые, с ровным краем. В жидких средах образуют равномерную светорассеивающую суспензию, при хранении без перемешивания оседают на дно. Клетки растут в интервале температур от 8°С до 43°С, интервал для наиболее эффективного культивирования - 28-38°С, оптимум роста при 37°С. Оптимальный интервал рН для культивирования рН 5-7.
Генетические признаки, устойчивость к антибиотикам: генотип исходного штамма Esherichia coli BL21(DE3)pLysS: F- отрТ hsdSB (rB- mB-) gal dcm (DE3) pLysS(CamR). Проявляет устойчивость к хлорамфениколу (34 мкг/мл), обусловленную наличием гена устойчивости в ДНК рекомбинантной плазмиды pLysS.
Характеристики полезного вещества, синтезируемого штаммом: рекомбинантный белок Cpf1, длиной 1281 аминокислотных остатков, состоящий из нуклеазы Cpf1 из бактериального штамма Moraxella bovis (1261 аминокислот) и вспомогательной последовательности длиной 20 аминокислот.
Продуктивность штамма - рекомбинантный Cpf1 составляет не менее 21 масс % по массе белка клеточного лизата при культивировании в жидкой среде ТВ (питательная среда TerrificBroth) при 37°С, 200 об/мин, в условиях индукции 1М ИПТГ (1:100) с добавлением антибиотиков хлорамфеникола (34 мкг/мл), ампициллина (100 мкг/мл) и рифампицина (100 мкг/мл).
Культивирование штамма: культивирование при температуре 37°С в термостате или качалке, в агаризованной (2% агара) LB или жидкой ТВ-среде соответственно.
Селективные условия - в культуральную среду добавлены 100 мкг/мл ампициллина и 34 мкг/мл хлорамфеникола.
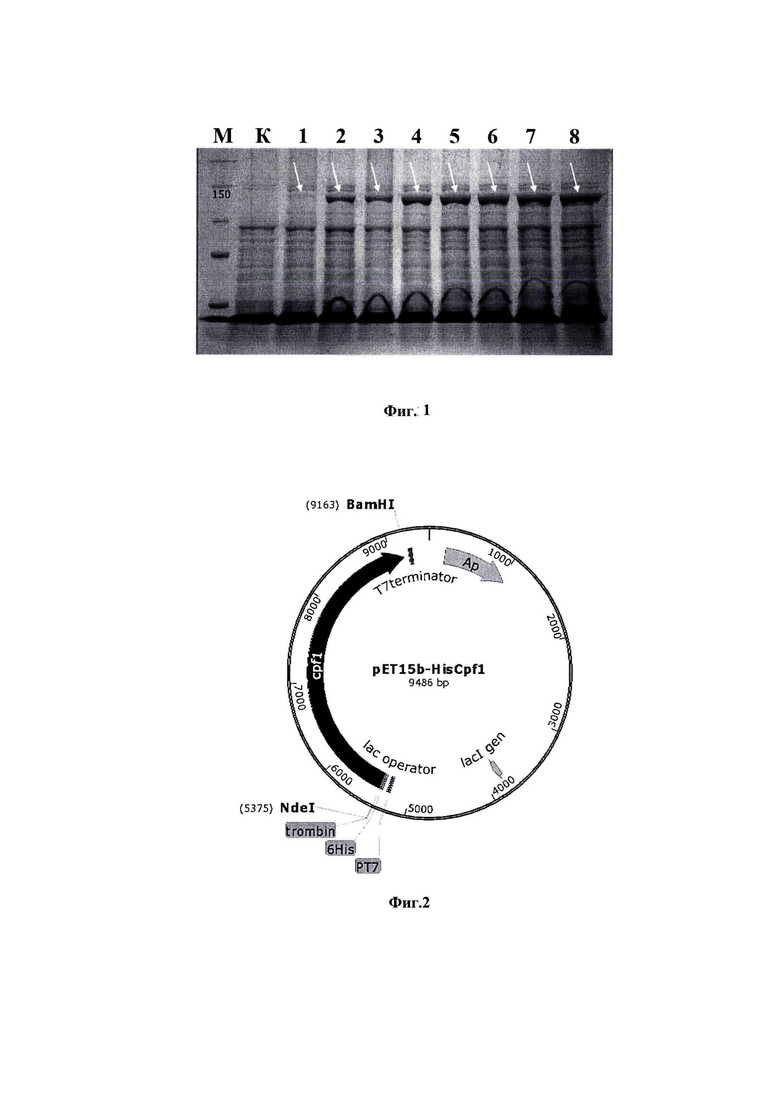
На фиг. 1 приведены электрофореграммы лизатов клеток E. coli BL21(DE3)pLysS/pET15b-HisCpf1 при культивировании в условиях индукции синтеза белка при помощи ИПТГ и без индукции, где М - белковый маркер PrecisionPlusProtein™ Unstained Protein Standards (Bio-rad); К - контрольная неиндуцированная культура, 1-8-1-8 ч после индукции 1М ИПТГ (1:100) соответственно.
Сущность изобретения поясняется следующими конкретными примерами получения и использования штамма E. coli BL21(DE3)pLysS/pET15b-HisCpf1.
Пример 1. Создание генетической конструкции, обеспечивающей синтез рекомбинантной нуклеазы Cpf1 в клетках E. coli.
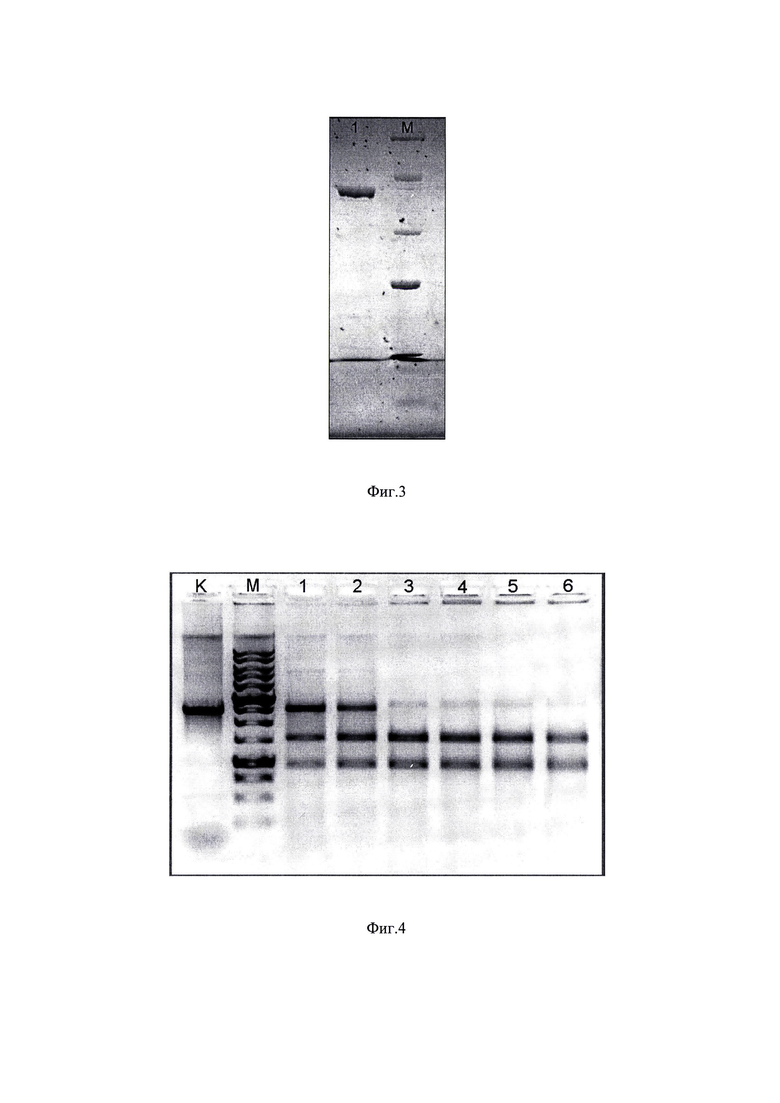
Методом полимеразной цепной реакции с использованием специфических праймеров Cpf1-For (SEQ ID NO 1) и Cpf1-Rev (SEQ ID NO 2) на матрице геномной ДНК штамма Moraxella bovis был амплифицирован ген cpf1 Последовательность нуклеотидов полученного гена совпадает с последовательностью cpf1 Moraxella bovis, имеющейся в GenBank (СР030241.1) Полученный ген был встроен в экспрессирующий вектор pET15b с получением экспрессионной плазмиды pET15b-HisCpf1 (фиг. 2).
Плазмида pET15b-HisCpf1 обеспечивает в клетках Е. coli синтез полноразмерной бактериальной нуклеазы, дополнительно содержащей с N-конца вспомогательную последовательность длиной 20 аминокислот, которая содержит участок, состоящий из 6-ти остатков гистидина (гистидиновая метка), предназначенный для последующей очистки рекомбинантной нуклеазы с помощью металлохелатной хроматографии, а также сайт гидролиза тромбином (протеиназой), позволяющий при необходимости удалить большую часть служебной последовательности при обработке очищенного препарата рекомбинантной нуклеазы тромбином. Нуклеотидная (SEQ ID NO 4) и аминокислотная (SEQ ID NO 5) последовательности рекомбинантной нуклеазы Cpf1 представлены в Приложении 1.
Пример 2. Получение штамма-продуцента рекомбинантной нуклеазы Cpf1 и исследование его продуктивности.
Полученной плазмидой pET15b-HisCpf1 были трансформированы клетки Е. coli штамма BL21(DE3)pLysS, содержащие в своем геноме ген, кодирующий полимеразу фага Т7 под контролем бактериального промотора, индуцируемого лактозой или ИПТГ. Также данный штамм несет плазмиду pLysS, кодирующую ингибитор Т7 полимеразы - лизоцим фага Т7, что существенно снижает базальный уровень экспрессии гена, встроенного под Т7 промотор, что позволяет клонировать даже токсичные для клетки гены. Кроме того, клетки Е. coli BL21(DE3)pLysS дефектны по генам протеаз Ion и ompT. Отсутствие этих двух протеаз уменьшает деградацию гетерологичных белков.
В результате был получен штамм E. coli BL21(DE3)pLysS/pET15b-HisCpf1- продуцент бактериального белка Cpf1.
Для поддержания полученного штамма-продуцента белка Cpf1 использовали плотную агаризованную LB-среду, содержащую 100 мкг/мл ампициллина и 34 мкг/мл хлорамфеникола.
Продуктивность полученного штамма-продуцента изучали путем культивирования клеток в среде ТВ, в термостатированном шейкере роторного типа при температуре 37°С, скорости вращения платформы 200 об/мин. По достижении культурой оптической плотности 3 о.е. индуцировалась экспрессия целевого гена (добавление 1М ИПТГ (1:100), а затем добавление рифампицина до концентрации 100 мкг/мл через 30 минут после начала индукции). При добавлении рифампицина происходит нарушение экспрессии генов домашнего хозяйства, поскольку рифампицин ингибирует ДНК-зависимую РНК-полимеразу у Е. coli, при этом экспрессия целевого белка не нарушается, так как Т7 РНК-полимераза, под промотором которой находится целевой ген, устойчива к действию рифампицина. В качестве контроля использовали неиндуцированную культуру (без добавления ИПТГ). Образцы собранной центрифугированием биомассы клеток лизировали и анализировали методом электрофореза в полиакриламидном геле в денатурирующих условиях. Результат представлен на фиг. 1.
Как видно из фиг. 1, индукция ИПТГ культуры клеток Е. coli BL21(DE3)pLysS/pET 15b-HisCpf1 приводит к синтезу белка с молекулярным весом примерно 147 кДа, что соответствует ожидаемому молекулярному весу для рекомбинантного Cpf1. Анализ денситограммы полиакриламидного геля, представленного на фиг. 1, выполненный с помощью программы CLIQS, показал, что рекомбинантный Cpf1 составляет 21% общего белка клеточного лизата при начальной оптической плотности 3 о.е. и инкубации с ИПТГ и рифампицином в течение 5 часов.
Пример 3. Очистка рекомбинантного Cpf1 и изучение его биологической активности.
Рекомбинантный Cpf1 очищали из клеточных лизатов методом металлохелатной хроматографии с последующим концентрированием белковой фракции сульфатом аммония и обессоливанием при помощи спин-колонок. Результат очистки клеточного лизата Е. coli BL21(DE3)pLysS/pET15b-HisCpf1 представлен на фиг. 3, где М - белковый маркер PrecisionPlusProtein™ UnstainedProteinStandards (Bio-rad), 1 - очищенный препарат Cpf1.
Как видно из фиг. 3 препарат Cpf1 является практически гомогенным и его чистота составляет не менее 90% по результатам электрофореза в полиакриламидном геле с последующей денситометрией.
Биологическая активность рекомбинантного Cpf1 оценивалась in vitro, матрицей для гидролиза был выбран фрагмент плазмиды pUC19, линеаризованной при помощи ПЦР. Для постановки гидролиза реакционная смесь, состоящая из буферного раствора, ионов магния, очищенного препарата Cpf1 и gPHK (SEQ ID NO 3), инкубировалась в течение 10 минут при комнатной температуре для образования комплексов фермент-РНК. Далее к реакционной смеси добавляли линеаризованный фрагмент pUC19, смесь инкубировали 10 минут при 37°С в твердотельном термостате. Результаты анализировали в 1% агарозном геле. Результаты экспериментов по определению биологической активности рекомбинантного Cpf1 представлены на фиг. 4, где М - ДНК-маркер 1 кб (СибЭнзим), К - контрольная исходная матрица pUC19, 1-3 - препараты ДНК после гидролиза, соотношение ДНК:РНК/Cpf1 = 1:1, 1:2, 1:3 соответственно.
Из фиг. 4 следует, что рекомбинантный Cpf1, продуцируемый клетками штамма Е. coli ВЕ21(ОЕ3)pLysS/pET15b1Cpf1, обладает биологической активностью и при мольном соотношении ДНК:РНК:Cpf1=1:3 гидролиз протекает на 100%.
Использованные источники информации
1. Zetsche B. и др. Cpf1 Is a Single RNA-Guided Endonuclease of a Class 2 CRISPR-Cas System // Cell. 2015. T. 163, №3. C. 759-771.
2. Shmakov S. и др. Discovery and Functional Characterization of Diverse Class 2 CRISPR-Cas Systems // Mol. Cell. NIH Public Access, 2015. T. 60, №3. C. 385-397.
--->
ПРИЛОЖЕНИЕ 1
<120> Штамм Escherichia coli BL21(DE3)pLysS/pET15b-HisCpf1 – продуцент
РНК-направляемой эндонуклеазы CRISPR/Cpf1
<160> 5
<210> 1
<211> 40
<212> DNA
<213> Искусственная последовательность
<400> 1
Cgcgcggcagccatatgttatttcaagagtttacccattt 40
<210> 2
<211> 35
<212> DNA
<213> Искусственная последовательность
<400> 2
Gttagcagccggatcttagcggttttgagcgaagt 35
<210> 3
<211> 43
<212> RNA
<213> Искусственная последовательность
<400> 3
Aaauuucuacuguuuguagaucucacauguucuuuccugcguu 43
<210> 4
<211> 43
<212> DNA
<213> Искусственная последовательность
<400> 4
atgttatttc aagagtttac ccatttatat cccctatcaa aaaccgtgcg ttttgaatta 60
aagcccattg gcaagacatt agagcatatc catgccaaaa actttttgag ccaagatgag 120
accatggcgg acatgtacca aaaggtgaag gcgatgttgg acgattatca tcgtgatttt 180
atcgctgata tgatgggcga agttaagcta actaagctgg cagaatttta tgatgtgtat 240
ttaaaattta gaaaaaatcc caaagacgat ggattgcaaa aacagctaaa ggacttgcaa 300
gcagttttaa gaaaagagat tgtgaagccc atcggtaatg gcggaaaata caaggcgggt 360
tatgaccgct tgtttggggc aaagctcttt aaggacggca agaagcttgg tgatttggcg 420
aaatttgtca tcgcccaaga gggcgactca tcacccaagc ttgctcacct tgcccatttt 480
gagaagttta gcacttactt tactggcttt catgacaacc gcaaaaacat gtacagcgat 540
gaggataaac acacctccat tgcctatcgt ctgattcatg aaaacttacc acgctttatt 600
gacaacctac aaatactgac aaccatcaaa caaaagcact cggcgttgta tgaccagatt 660
ataaatgagc tgactgcgag tggcttggat gtgtcgctgg caagtcatct ggacggctat 720
cacaagcttt taacccaaga gggtatcacg gcttataata ccctgcttgg cggtatcagt 780
ggcgaggcag gttctcgcaa gataaaaggt atcaatgagc ttatcaatag tcatcacaac 840
caacattgcc acaaatccga acgtatcgcc aaattacgcc ccctgcacaa acaaatccta 900
agcgacggca tgggtgtgtc atttttgcca agcaaatttg ctgatgacag cgaagtttgt 960
caagcggtca atgaatttta ccgccattat gctgatgttt ttgccaaggt gcaaagctta 1020
tttgatgggt ttgatgacta tcaaaaggac ggcatttatg ttgagcataa aaatttaaat 1080
gaactatcca agcaggcatt tggtgatttt gccttgttgg gtcgggtgtt agacggctat 1140
tatgtggatg tggtgagtcc agagtttaat gagagatttg ccaaagccaa aaccgacaat 1200
gccaaagaaa aactaacaaa agaaaaagac aaattcatca aaggcgtgca ttccttggca 1260
agccttgagc aggcgataga gcattatact gctaggcatg atgatgagtc tgtgcaggcg 1320
ggcaaacttg gacagtattt caaacacggt ctggcaggcg tggacaatcc aatccaaaaa 1380
atacacaaca accacagcac cattaagggt tttttggagc gtgaacgtcc agcaggcgag 1440
cgagcattgc ccaaaattca gttaggcaaa aaccctgaaa tcaggcaact aaaagagctg 1500
ctagacaaca ccctaaacgt ggtgcatttt gccaagttat taacgaccaa aaccacgcta 1560
gacaaccaag acggcaattt ttatggtgag tttggggcgt tgtatgatga gcttgccaag 1620
attcccacgc tttataacaa agtgcgtgat tatctgtcgc aaaagccatt tagcaccgaa 1680
aaatataaat taaactttgg caatccgaca ttattaaacg ggtgggattt gaataaagaa 1740
aaagataatt ttggggttat cttacaaaaa gacggttgtt attatttggc gttattagat 1800
aaagctcata aaaaagtatt tgataatgct ccaaatacag gcaaaaatat ttatcaaaaa 1860
atgatttata aattattgcc cggacctaat aaaatgctac ccaaagtatt ttttgccaag 1920
agtaatttgg attattataa cccttctgct gaactactgg ataaatatgc aaaaggcacg 1980
cacaaaaaag gcgataattt taatctaaaa gactgccatg cgttgattga tttttttaag 2040
gcgggtatta ataaacatcc agaatggcaa cattttggtt ttaaattttc gccaaccagt 2100
agctatcagg atttgagtga tttttataga gaagtcgaac cacaaggtta tcaagtaaaa 2160
tttgtggata tcaacgccga ttatattgac gagttggttg aacaaggtca gttgtattta 2220
ttccagattt ataataaaga tttctcgccc aaggctcacg gtaagcccaa tttgcatact 2280
ctgtatttta aggcgttgtt tagcgaagat aaccttgccg atccaattta taaattaaat 2340
ggcgaggcac agatatttta tcgtaaggca tctttggata tgaatgaaac caccattcat 2400
cgtgcaggcg aggtattaga aaataaaaat cctgataatc caaaaaagcg tcaatttgtc 2460
tatgacatca tcaaagataa acgctatacc caagataagt ttatgctaca tgtacctatt 2520
accatgaatt ttggtgtgca gggcatgacg attaaagaat ttaataaaaa agttaatgaa 2580
agcatacagc aatatgatga ggtgaatgtc ataggcatag accgtggaga gcgacatttg 2640
ctatatctga ccgtgattaa tagcaaaggc gaaatcttag aacagcgtag tctcaatgac 2700
atcatcacca catcggcaaa tggtacacaa atgaccacgc attatcataa aatactagac 2760
aagagagaaa tagaacgcct aaatgctcgt gtcggttggg gtgagattga gaccataaaa 2820
gagctaaaat cgggctatct aagccatgtg gtacatcaaa tcagccagct tatgcttaaa 2880
tataatgcca ttgtggtgtt agaggattta aattttgggt ttaaacgtgg tcgctttaag 2940
gtggaaaaac aaatttacca aaactttgaa aacgccttaa tcaaaaagct aaaccatttg 3000
gtattaaaag ataaggcaga tgatgagatt ggctcataca aaaatgccct gcaactgacc 3060
aataatttta ctgatctaaa aagcattggc aaacaaacag gctttttatt ctatgtgcct 3120
gcatggaata ccagtaaaat agaccctgaa acgggctttg tggatttgtt aaaaccacgt 3180
tatgaaaata tcgctcagtc gcaagcgttt tttggtaaat ttgataagat ttgttataac 3240
gcagataagg gttattttga atttcatatt gattatgcca aatttactga caaagccaaa 3300
aactctcgcc aaaaatggac gatttgctcg catggcgata aacgttatgt gtatgacaaa 3360
accgccaata aaaataaagg aacgacaaag ggtattaatg tcaatgatga attaaaatcg 3420
ctatttgctc gtcatcacat caatgaaaaa cagccaaatc tggtcatgga tatttgccaa 3480
aataacgata aagaatttca taaatcgctg atgtatctat taaagacatt gcttgcatta 3540
agatatagta acgccagtag cgatgaagat tttattttat cgcctgtggc aaatgatgag 3600
ggtgtgtttt ttaattcggc attggcggat gatacacagc cacaaaatgc ggacgccaat 3660
ggggcgtatc atatcgcatt aaagggctta tggctattaa atgaactaaa agacagcgat 3720
gatttgaata aagtcaaact tgccattgac aatcaaacat ggctaaactt cgctcaaaac 3780
cgctaa 3786
<210> 5
<211> 43
<212> PRT
<213> Искусственная последовательность
<400> 5
Met Leu Phe Gln Glu Phe Thr His Leu Tyr Pro Leu Ser Lys Thr Val
1 5 10 15
Arg Phe Glu Leu Lys Pro Ile Gly Lys Thr Leu Glu His Ile His Ala
20 25 30
Lys Asn Phe Leu Ser Gln Asp Glu Thr Met Ala Asp Met Tyr Gln Lys
35 40 45
Val Lys Ala Met Leu Asp Asp Tyr His Arg Asp Phe Ile Ala Asp Met
50 55 60
Met Gly Glu Val Lys Leu Thr Lys Leu Ala Glu Phe Tyr Asp Val Tyr
65 70 75 80
Leu Lys Phe Arg Lys Asn Pro Lys Asp Asp Gly Leu Gln Lys Gln Leu
85 90 95
Lys Asp Leu Gln Ala Val Leu Arg Lys Glu Ile Val Lys Pro Ile Gly
100 105 110
Asn Gly Gly Lys Tyr Lys Ala Gly Tyr Asp Arg Leu Phe Gly Ala Lys
115 120 125
Leu Phe Lys Asp Gly Lys Lys Leu Gly Asp Leu Ala Lys Phe Val Ile
130 135 140
Ala Gln Glu Gly Asp Ser Ser Pro Lys Leu Ala His Leu Ala His Phe
145 150 155 160
Glu Lys Phe Ser Thr Tyr Phe Thr Gly Phe His Asp Asn Arg Lys Asn
165 170 175
Met Tyr Ser Asp Glu Asp Lys His Thr Ser Ile Ala Tyr Arg Leu Ile
180 185 190
His Glu Asn Leu Pro Arg Phe Ile Asp Asn Leu Gln Ile Leu Thr Thr
195 200 205
Ile Lys Gln Lys His Ser Ala Leu Tyr Asp Gln Ile Ile Asn Glu Leu
210 215 220
Thr Ala Ser Gly Leu Asp Val Ser Leu Ala Ser His Leu Asp Gly Tyr
225 230 235 240
His Lys Leu Leu Thr Gln Glu Gly Ile Thr Ala Tyr Asn Thr Leu Leu
245 250 255
Gly Gly Ile Ser Gly Glu Ala Gly Ser Arg Lys Ile Lys Gly Ile Asn
260 265 270
Glu Leu Ile Asn Ser His His Asn Gln His Cys His Lys Ser Glu Arg
275 280 285
Ile Ala Lys Leu Arg Pro Leu His Lys Gln Ile Leu Ser Asp Gly Met
290 295 300
Gly Val Ser Phe Leu Pro Ser Lys Phe Ala Asp Asp Ser Glu Val Cys
305 310 315 320
Gln Ala Val Asn Glu Phe Tyr Arg His Tyr Ala Asp Val Phe Ala Lys
325 330 335
Val Gln Ser Leu Phe Asp Gly Phe Asp Asp Tyr Gln Lys Asp Gly Ile
340 345 350
Tyr Val Glu His Lys Asn Leu Asn Glu Leu Ser Lys Gln Ala Phe Gly
355 360 365
Asp Phe Ala Leu Leu Gly Arg Val Leu Asp Gly Tyr Tyr Val Asp Val
370 375 380
Val Ser Pro Glu Phe Asn Glu Arg Phe Ala Lys Ala Lys Thr Asp Asn
385 390 395 400
Ala Lys Glu Lys Leu Thr Lys Glu Lys Asp Lys Phe Ile Lys Gly Val
405 410 415
His Ser Leu Ala Ser Leu Glu Gln Ala Ile Glu His Tyr Thr Ala Arg
420 425 430
His Asp Asp Glu Ser Val Gln Ala Gly Lys Leu Gly Gln Tyr Phe Lys
435 440 445
His Gly Leu Ala Gly Val Asp Asn Pro Ile Gln Lys Ile His Asn Asn
450 455 460
His Ser Thr Ile Lys Gly Phe Leu Glu Arg Glu Arg Pro Ala Gly Glu
465 470 475 480
Arg Ala Leu Pro Lys Ile Gln Leu Gly Lys Asn Pro Glu Ile Arg Gln
485 490 495
Leu Lys Glu Leu Leu Asp Asn Thr Leu Asn Val Val His Phe Ala Lys
500 505 510
Leu Leu Thr Thr Lys Thr Thr Leu Asp Asn Gln Asp Gly Asn Phe Tyr
515 520 525
Gly Glu Phe Gly Ala Leu Tyr Asp Glu Leu Ala Lys Ile Pro Thr Leu
530 535 540
Tyr Asn Lys Val Arg Asp Tyr Leu Ser Gln Lys Pro Phe Ser Thr Glu
545 550 555 560
Lys Tyr Lys Leu Asn Phe Gly Asn Pro Thr Leu Leu Asn Gly Trp Asp
565 570 575
Leu Asn Lys Glu Lys Asp Asn Phe Gly Val Ile Leu Gln Lys Asp Gly
580 585 590
Cys Tyr Tyr Leu Ala Leu Leu Asp Lys Ala His Lys Lys Val Phe Asp
595 600 605
Asn Ala Pro Asn Thr Gly Lys Asn Ile Tyr Gln Lys Met Ile Tyr Lys
610 615 620
Leu Leu Pro Gly Pro Asn Lys Met Leu Pro Lys Val Phe Phe Ala Lys
625 630 635 640
Ser Asn Leu Asp Tyr Tyr Asn Pro Ser Ala Glu Leu Leu Asp Lys Tyr
645 650 655
Ala Lys Gly Thr His Lys Lys Gly Asp Asn Phe Asn Leu Lys Asp Cys
660 665 670
His Ala Leu Ile Asp Phe Phe Lys Ala Gly Ile Asn Lys His Pro Glu
675 680 685
Trp Gln His Phe Gly Phe Lys Phe Ser Pro Thr Ser Ser Tyr Gln Asp
690 695 700
Leu Ser Asp Phe Tyr Arg Glu Val Glu Pro Gln Gly Tyr Gln Val Lys
705 710 715 720
Phe Val Asp Ile Asn Ala Asp Tyr Ile Asp Glu Leu Val Glu Gln Gly
725 730 735
Gln Leu Tyr Leu Phe Gln Ile Tyr Asn Lys Asp Phe Ser Pro Lys Ala
740 745 750
His Gly Lys Pro Asn Leu His Thr Leu Tyr Phe Lys Ala Leu Phe Ser
755 760 765
Glu Asp Asn Leu Ala Asp Pro Ile Tyr Lys Leu Asn Gly Glu Ala Gln
770 775 780
Ile Phe Tyr Arg Lys Ala Ser Leu Asp Met Asn Glu Thr Thr Ile His
785 790 795 800
Arg Ala Gly Glu Val Leu Glu Asn Lys Asn Pro Asp Asn Pro Lys Lys
805 810 815
Arg Gln Phe Val Tyr Asp Ile Ile Lys Asp Lys Arg Tyr Thr Gln Asp
820 825 830
Lys Phe Met Leu His Val Pro Ile Thr Met Asn Phe Gly Val Gln Gly
835 840 845
Met Thr Ile Lys Glu Phe Asn Lys Lys Val Asn Glu Ser Ile Gln Gln
850 855 860
Tyr Asp Glu Val Asn Val Ile Gly Ile Asp Arg Gly Glu Arg His Leu
865 870 875 880
Leu Tyr Leu Thr Val Ile Asn Ser Lys Gly Glu Ile Leu Glu Gln Arg
885 890 895
Ser Leu Asn Asp Ile Ile Thr Thr Ser Ala Asn Gly Thr Gln Met Thr
900 905 910
Thr His Tyr His Lys Ile Leu Asp Lys Arg Glu Ile Glu Arg Leu Asn
915 920 925
Ala Arg Val Gly Trp Gly Glu Ile Glu Thr Ile Lys Glu Leu Lys Ser
930 935 940
Gly Tyr Leu Ser His Val Val His Gln Ile Ser Gln Leu Met Leu Lys
945 950 955 960
Tyr Asn Ala Ile Val Val Leu Glu Asp Leu Asn Phe Gly Phe Lys Arg
965 970 975
Gly Arg Phe Lys Val Glu Lys Gln Ile Tyr Gln Asn Phe Glu Asn Ala
980 985 990
Leu Ile Lys Lys Leu Asn His Leu Val Leu Lys Asp Lys Ala Asp Asp
995 1000 1005
Glu Ile Gly Ser Tyr Lys Asn Ala Leu Gln Leu Thr Asn Asn Phe Thr
1010 1015 1020
Asp Leu Lys Ser Ile Gly Lys Gln Thr Gly Phe Leu Phe Tyr Val Pro
1025 1030 1035 1040
Ala Trp Asn Thr Ser Lys Ile Asp Pro Glu Thr Gly Phe Val Asp Leu
1045 1050 1055
Leu Lys Pro Arg Tyr Glu Asn Ile Ala Gln Ser Gln Ala Phe Phe Gly
1060 1065 1070
Lys Phe Asp Lys Ile Cys Tyr Asn Ala Asp Lys Gly Tyr Phe Glu Phe
1075 1080 1085
His Ile Asp Tyr Ala Lys Phe Thr Asp Lys Ala Lys Asn Ser Arg Gln
1090 1095 1100
Lys Trp Thr Ile Cys Ser His Gly Asp Lys Arg Tyr Val Tyr Asp Lys
1105 1110 1115 1120
Thr Ala Asn Lys Asn Lys Gly Thr Thr Lys Gly Ile Asn Val Asn Asp
1125 1130 1135
Glu Leu Lys Ser Leu Phe Ala Arg His His Ile Asn Glu Lys Gln Pro
1140 1145 1150
Asn Leu Val Met Asp Ile Cys Gln Asn Asn Asp Lys Glu Phe His Lys
1155 1160 1165
Ser Leu Met Tyr Leu Leu Lys Thr Leu Leu Ala Leu Arg Tyr Ser Asn
1170 1175 1180
Ala Ser Ser Asp Glu Asp Phe Ile Leu Ser Pro Val Ala Asn Asp Glu
1185 1190 1195 1200
Gly Val Phe Phe Asn Ser Ala Leu Ala Asp Asp Thr Gln Pro Gln Asn
1205 1210 1215
Ala Asp Ala Asn Gly Ala Tyr His Ile Ala Leu Lys Gly Leu Trp Leu
1220 1225 1230
Leu Asn Glu Leu Lys Asp Ser Asp Asp Leu Asn Lys Val Lys Leu Ala
1235 1240 1245
Ile Asp Asn Gln Thr Trp Leu Asn Phe Ala Gln Asn Arg
1250 1255 1260
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| МОДИФИЦИРОВАННЫЕ МИТОХОНДРИИ И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2811467C2 |
| НОВАЯ ПСИКОЗО-6-ФОСФАТ ФОСФАТАЗА, КОМПОЗИЦИЯ ДЛЯ ПОЛУЧЕНИЯ ПСИКОЗЫ, СОДЕРЖАЩАЯ УКАЗАННЫЙ ФЕРМЕНТ, СПОСОБ ПОЛУЧЕНИЯ ПСИКОЗЫ С ИСПОЛЬЗОВАНИЕМ УКАЗАННОГО ФЕРМЕНТА | 2018 |
|
RU2757229C2 |
| Способ получения рекомбинантного белка нуклеазы бактерий Serratia marcescens для гидролиза нуклеиновых кислот | 2021 |
|
RU2773954C1 |
| Рекомбинантный белок нуклеазы бактерий Serratia marcescens для гидролиза нуклеиновых кислот | 2022 |
|
RU2787186C1 |
| Последовательность нуклеотидов, кодирующая β-N-ацетилгексозаминидазу Aggregatibacter actinomycetemcomitans, и генетические конструкции, её содержащие | 2021 |
|
RU2789544C1 |
| КОМПОЗИЦИЯ ПО ПРЕДУПРЕЖДЕНИЮ ИНФЕКЦИИ Mycoplasma spp. | 2013 |
|
RU2646137C2 |
| Рекомбинантная плазмидная ДНК pERIG-PGS, кодирующая гибридный белок, способный к автокаталитическому расщеплению с образованием антиангиогенного пептида пигастина - производного фрагмента [44-77] фактора роста пигментного эпителия человека, штамм Escherichia coli BL21(DE3)/pERIG-PGS - продуцент указанного белка, и способ получения рекомбинантного антиангиогенного пептида | 2017 |
|
RU2664199C1 |
| Рекомбинантный слитый белок | 2019 |
|
RU2732795C1 |
| НОВАЯ D-ПСИКОЗО-3-ЭПИМЕРАЗА И СПОСОБ ПОЛУЧЕНИЯ D-ПСИКОЗЫ С ЕЕ ИСПОЛЬЗОВАНИЕМ | 2017 |
|
RU2727903C1 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2737537C2 |
Изобретение относится к штамму Escherichia coli, продуцирующему рнк-направляемую эндонуклеазу CRISPR/CPF1. Предложен штамм Escherichia coli BL21(DE3)pLysS/pET15b-HisCpf1, продуцирующий рнк-направляемую эндонуклеазу CRISPR/CPF1 и полученный путем трансформации клеток Escherichia coli штамма BL21(DE3)pLysS рекомбинантной плазмидной ДНК рЕТ15b-HisCpf1, сконструированной на основе вектора pET15b, несущего ген рекомбинантного белка Cpf1 длиной 1281 аминокислотных остатков, состоящего из нуклеазы из бактериального штамма Moraxella bovis (1261 аминокислота) и вспомогательной последовательности длиной в 20 аминокислот, включающей в себя гистидиновую метку и сайт гидролиза для тромбина. Изобретение обеспечивает высокоэффективную продукцию активного рекомбинатного фермента Cpf1. 4 ил., 3 пр.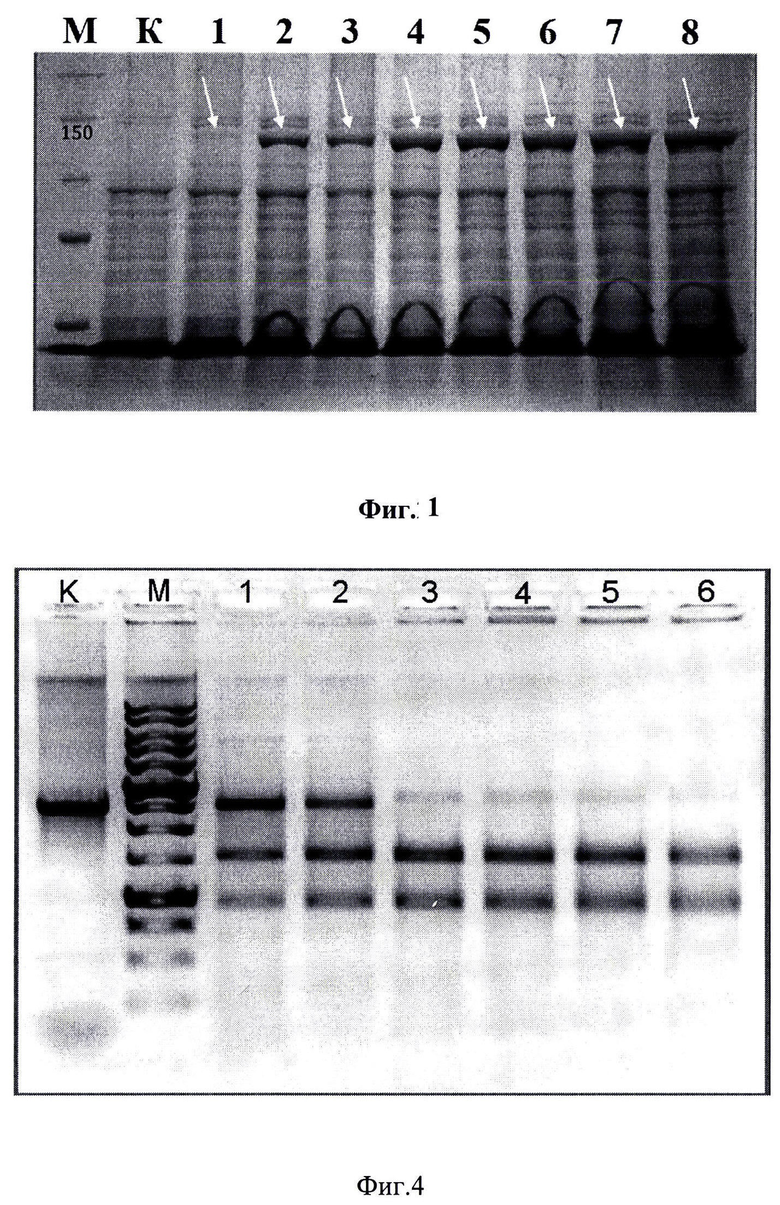
Штамм Escherichia coli BL21(DE3)pLysS/pET15b-HisCpf1 - продуцент рнк-направляемой эндонуклеазы CRISPR/CPF1, полученный путем трансформации клеток Escherichia coli штамма BL21(DE3)pLysS рекомбинантной плазмидной ДНК рЕТ15b-HisCpf1, сконструированной на основе вектора pET15b, несущего ген рекомбинантного белка Cpf1 длиной 1281 аминокислотных остатков, состоящего из нуклеазы из бактериального штамма Moraxella bovis (1261 аминокислота) и вспомогательной последовательности длиной в 20 аминокислот, включающей в себя гистидиновую метку и сайт гидролиза для тромбина.
| MOHANRAJU P | |||
| ET AL | |||
| Способ гальванического снятия позолоты с серебряных изделий без заметного изменения их формы | 1923 |
|
SU12A1 |
| Bio Protoc | |||
| Способ получения цианистых соединений | 1924 |
|
SU2018A1 |
| Способ получения цианистых соединений | 1924 |
|
SU2018A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Найдено онлайн: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8275269/ Дата обращения 14.01.2022 | |||
| ZETSCHE B | |||
| ET AL | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
Авторы
Даты
2022-06-15—Публикация
2021-07-05—Подача