Изобретение относится к молекулярной биотехнологии, вирусологии и эпидемиологии и может быть использовано в молекулярно-эпидемиологических исследованиях энтеровирусной инфекции, являющихся важным компонентом эпидемиологического надзора, а также в научных исследованиях с целью оптимизации филогенетического анализа штаммов энтеровируса Коксаки А10.
Актуальность энтеровирусной инфекции (ЭВИ) для населения Российской Федерации (РФ) обусловлена возникновением ежегодных сезонных подъемов заболеваемости с формированием вспышечных очагов. При этом информативные молекулярно-эпидемиологические исследования являются неотъемлемой составляющей эффективного надзора за этой распространенной инфекцией [1].
В настоящее время известно более 100 типов энтеровирусов, инфицирующих человека. Одним из наиболее значимых типов энтеровирусов является Коксаки А10, который в последнее десятилетие, наряду с энтеровирусом Коксаки А6, стал чаще выявляться при вспышках ЭВИ [2].
Большинство имеющихся в международной базе данных GenBank (National Center for Biotechnological Information, NCBI) нуклеотидных последовательностей энтеровируса Коксаки А10 представлено короткими фрагментами генома, не позволяющими в полной мере изучить молекулярно-эпидемиологические особенности циркуляции этого вируса. Получение же нуклеотидной последовательности целого генома энтеровируса или полных последовательностей наиболее консервативных его участков, в частности гена VP1, при проведении филогенетического анализа дает более точное представление о происхождении вируса, что является особенно важным при расследовании крупных вспышек ЭВИ, помогает выявить наличие генетических вариантов одного типа энтеровируса, в том числе одновременное их присутствие на одной территории.
В научной литературе представлен способ получения полной нуклеотидной последовательности гена VP1 энтеровирусов вида А: «Видоспецифическая амплификация энтеровирусов человека методом ОТ-ПЦР: способ быстрой идентификации вида не охарактеризованных энтеровирусов» (Oberste MS, Maher K, Williams AJ, Dybdahl-Sissoko N, Brown BA, Gookin MS,  S, Mishrik N, Uddin M, Pallansch MA. Species-specific RT-PCR amplification of human enteroviruses: a tool for rapid species identification of uncharacterized enteroviruses. J Gen Virol. 2006; 87: 119-128).
S, Mishrik N, Uddin M, Pallansch MA. Species-specific RT-PCR amplification of human enteroviruses: a tool for rapid species identification of uncharacterized enteroviruses. J Gen Virol. 2006; 87: 119-128).
К недостатку данного способа можно отнести то, что представленные в данной работе пары праймеров для энтеровирусов вида А (486/488 и 487/489) являются не специфичными по отношению к последовательности гена VP1 энтеровируса Коксаки А10.
Задачей разработки является определение синтетических олигонуклеотидов (праймеров) для амплификации перекрывающихся фрагментов генома, позволяющих получить полную нуклеотидную последовательность гена VP1 энтеровируса Коксаки А10.
Технический результат заключается в обеспечении проведения оптимального и информативного филогенетического анализа штаммов Коксаки А10.
Указанный технический результат достигается разработкой набора синтетических олигонуклеотидных праймеров для получения полной нуклеотидной последовательности гена VP1 энтеровируса Коксаки А10, содержащего две пары олигонуклеотидов, специфичных к гену VP1 энтеровируса Коксаки А10, обладающих активностью в полимеразной цепной реакции, имеющие следующую структуру:
1) Прямой: 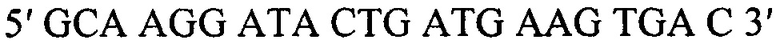
Обратный: 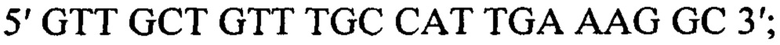
2) Прямой: 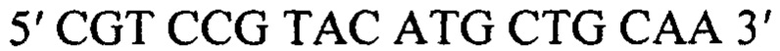
Обратный: 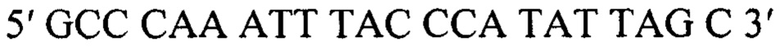
Для разработки предлагаемого к патентованию набора синтетических олигонуклеотидных праймеров проанализированы полные геномы энтеровируса Коксаки А10, депонированные в GenBank с 2008 по 2018 гг., в результате чего были выбраны наиболее консервативные участки на границах областей VP3-VP1 и VP1-2А генома Коксаки А10, что позволило подобрать две пары олигонуклеотидных праймеров (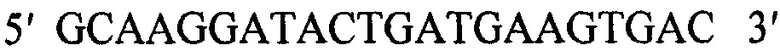 и
и 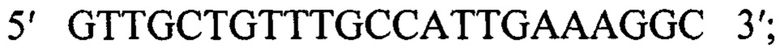
 и
и 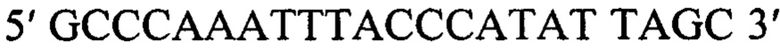 ), фланкирующие перекрывающиеся фрагменты, при последующей сборке которых после секвенирования удалось получить полные нуклеотидные последовательности гена VP1 штаммов энтеровируса Коксаки А10.
), фланкирующие перекрывающиеся фрагменты, при последующей сборке которых после секвенирования удалось получить полные нуклеотидные последовательности гена VP1 штаммов энтеровируса Коксаки А10.
Проверку эффективности заявляемого набора синтетических олигонуклеотидных праймеров проводили следующим образом.
Проведение полимеразной цепной реакции с использованием заявляемого набора синтетических олигонуклеотидных праймеров:
В качестве матрицы для получения полной нуклеотидной последовательности гена VP1 энтеровируса Коксаки А10 (длина = 894 нуклеотидных основания) использовали пробы кДНК, которые ранее нами были определены как принадлежащие к энтеровирусу Коксаки А10 по результатам секвенирования так называемого «типирующего» фрагмента VP1 (≈370 нуклеотидных оснований) по методике W. Allan Nix, et al. [3], а также фрагмента VP2 (≈368 нуклеотидных оснований) по методике D. Nasri, et al. [4]. Всего для проверки эффективности заявляемых олигонуклеотидов было использовано 117 проб кДНК Коксаки А10, полученных из клинического материала от пациентов с энтеровирусной инфекцией из различных субъектов Дальневосточного федерального округа в период с 2014 по 2018 гг. и коллекционированных на базе ФБУН Хабаровский НИИ эпидемиологии и микробиологии Роспотребнадзора.
С помощью предлагаемого к патентованию набора олигонуклеотидных праймеров были получены 117 полных нуклеотидных последовательностей гена VP1 (894 н.о.) энтеровируса Коксаки А10, что подтверждено путем сравнения полученных сиквенсов с референсными, представленными в международной базе генетических данных GenBank.
Условия проведения полимеразной цепной реакции оптимизировали по температуре отжига праймеров (Tm). Реакцию проводили с применением окрашенной реакционной смеси для проведения ПЦР анализа «ScreenMix-HS» (производство ЗАО «Евроген», Россия). Для того, чтобы получить полную последовательность гена VP1 энтеровируса Коксаки А10 длиной 894 н.о., использовали две пары праймеров, фланкирующие перекрывающиеся участки длиной 561 и 472 н.о. соответственно. Концентрация каждого из 4 праймеров составила 10 пмоль/мкл. Объем реакционной смеси равнялся 50 мкл, объем матрицы - 5 мкл.
Амплификацию проводили в термоциклере Mastercycler Nexus Gradient (производитель «Eppendorf», Германия) по следующей программе: 95°С - 5 мин.; затем 40 циклов: 95°С - 30 с., 50°С - 30 с., 72°С - 60 с.; далее заключительный этап 72°С - 1 мин. Анализ полученных продуктов ПЦР проводили в 1% агарозном геле. Искомые фрагменты вырезали из геля и очищали с помощью спин-колонок и реагентов, входящих в состав набора QIAquick Gel Extraction Kit (производство «QIAGEN», Германия). Секвенирование проводили на автоматическом генетическом анализаторе Applied Biosystems 3500 с набором реагентов «BigDye Terminator v3.1 Cycle Sequencing Kit (производство «Thermo Fisher Scientific Inc», США). Для соединения полученных перекрывающихся участков гена VP1 и их выравнивания использовали программное обеспечение MEGA 6.0. Подтверждение, что полученная полная нуклеотидная последовательность VP1 длиной 894 н.о. принадлежит энтеровирусу Коксаки А10 осуществляли с помощью онлайн-инструмента BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi). Филогенетический анализ нуклеотидных последовательностей со статистической обработкой данных выполняли с использованием байесовского алгоритма с методами Монте-Карло по схеме цепи Маркова, представленного в программном обеспечении BEAST v1.8.4.
Разработка набора синтетических олигонуклеотидных праймеров и его апробирование проводилось в лаборатории Дальневосточного регионального научно-методического центра по изучению энтеровирусных инфекций ФБУН Хабаровский НИИ эпидемиологии и микробиологии Роспотребнадзора.
Литература
1. Лукашев А.Н., Голицына Л.Н., Вакуленко Ю.А., Ахмадишина Л.В., Романенкова Н.И., Сапега Е.Ю. и др. Современные возможности и направления развития молекулярно-эпидемиологического мониторинга в надзоре за энтеровирусными инфекциями. Опыт Российской Федерации. Инфекция и иммунитет. 2018; 8 (4): 452-464.
2. Lu QB, Zhang ХА, Wo Y et al. Circulation of Coxsackievirus A10 and A6 in hand-foot-mouth disease in China, 2009-2011. PLoS One. 2012; 7(12):e52073.
3. Nix WA, Oberste MS, Pallansch MA. Sensitive, seminested PCR amplification of VP1 sequences for direct identification of all enterovirus serotypes from original clinical specimens. J Clin Microbiol. 2006; 44(8):2698-704.
4. Nasri D, Bouslama L, Omar S, et al. Typing of human enterovirus by partial sequencing of VP2. J Clin Microbiol. 2007; 45(8): 2370-2379.
Приложение
Перечень последовательностей к заявке на изобретение
№ 2020130428 от 15.09.2020 г.
SEQ ID NO 1: gcaaggatac tgatgaagtg ac
SEQ ID NO 2: gttgctgttt gccattgaaa ggc
SEQ ID NO 3: cgtccgtaca tgctgcaa
SEQ ID NO 4: gcccaaattt acccatatta gc
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ амплификации области VP1 генома энтеровирусов вида Enterovirus B | 2021 |
|
RU2774424C1 |
| Способ дифференциальной амплификации фрагмента области VP1 генома энтеровирусов видов Enterovirus A и Enterovirus B | 2020 |
|
RU2743352C1 |
| СПОСОБ ИДЕНТИФИКАЦИИ 5'-НТР ГЕНОМА ЭНТЕРОВИРУСОВ ГЕНОГРУППЫ ЭВI И ГЕНОГРУППЫ ЭВII С ИСПОЛЬЗОВАНИЕМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2010 |
|
RU2441917C2 |
| ШТАММ ЭНТЕРОВИРУСА А71 ТИПА СУБГЕНОТИПА С4, ИСПОЛЬЗУЕМЫЙ ДЛЯ ДИАГНОСТИКИ И ИЗУЧЕНИЯ ЭФФЕКТИВНОСТИ ЛЕЧЕБНО-ПРОФИЛАКТИЧЕСКИХ И ВАКЦИННЫХ ПРЕПАРАТОВ | 2014 |
|
RU2565811C1 |
| СПОСОБ ОБНАРУЖЕНИЯ И ДИФФЕРЕНЦИАЦИИ РНК ЭНТЕРОВИРУСОВ | 2000 |
|
RU2189396C2 |
| Способ генотипирования энтеровирусов методом секвенирования 1A-1B участка генома | 2019 |
|
RU2701145C1 |
| НАБОР ОЛИГОДЕЗОКСИРИБОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕННОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ РНК ЭНТЕРОВИРУСОВ МЕТОДОМ ОБРАТНОЙ ТРАНСКРИПЦИИ - ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ С ГИБРИДИЗАЦИОННО-ФЛУОРЕСЦЕНТНОЙ ДЕТЕКЦИЕЙ | 2011 |
|
RU2459830C1 |
| ШТАММ ЭНТЕРОВИРУСА КОКСАКИ В6, СЕЛЕКТИВНО ИНФИЦИРУЮЩИЙ И ЛИЗИРУЮЩИЙ ОПУХОЛЕВЫЕ КЛЕТКИ ЧЕЛОВЕКА in vitro | 2012 |
|
RU2496873C1 |
| Способ обнаружения генетического материала энтеровируса D68 методом полимеразной цепной реакции в реальном времени | 2023 |
|
RU2828885C1 |
| Способ получения вирусоподобных частиц норовируса, содержащих белок VP1 энтеровируса Echovirus 30, in vitro | 2022 |
|
RU2789354C1 |
Изобретение относится к области биотехнологии. Изобретение представляет собой набор синтетических олигонуклеотидных праймеров для получения полной нуклеотидной последовательности гена VP1 энтеровируса Коксаки А10, содержащего две пары олигонуклеотидов, специфичных к гену VP1 энтеровируса Коксаки А10, обладающих активностью в полимеразной цепной реакции, имеющих следующую структуру: 1) Прямой: 5' GCAAGGATACTGATGAAGTGAC 3' Обратный: 5' GTTGCTGTTTGCCATTGAAAGGC 3'; 2) Прямой: 5' CGTCCGTACATGCTGCAA 3' Обратный: 5' GCCCAAATTTACCCATATTAGC 3'. Изобретение позволяет обеспечить проведение оптимального и информативного филогенетического анализа штаммов Коксаки А10.
Набор синтетических олигонуклеотидных праймеров для получения полной нуклеотидной последовательности гена VP1 энтеровируса Коксаки А10, содержащий две пары олигонуклеотидов, специфичных к последовательности гена VP1 энтеровируса Коксаки А10, обладающих активностью в полимеразной цепной реакции, имеющих следующую структуру:
1) Прямой: 5' GCAAGGATACTGATGAAGTGAC 3'
Обратный: 5' GTTGCTGTTTGCCATTGAAAGGC 3';
2) Прямой: 5' CGTCCGTACATGCTGCAA 3'
Обратный: 5' GCCCAAATTTACCCATATTAGC 3'.
| НАБОР ОЛИГОДЕЗОКСИРИБОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНЫХ ДНК-ЗОНДОВ ДЛЯ ИДЕНТИФИКАЦИИ РНК ЭНТЕРОВИРУСОВ, РИНОВИРУСОВ, ВИРУСОВ ГЕПАТИТА А И Е ИЗ ВОДНОЙ СРЕДЫ МЕТОДОМ МУЛЬТИПЛЕКСНОЙ ПЦР | 2013 |
|
RU2542968C2 |
| ЛУКАШЕВ А.Н | |||
| и др | |||
| Современные возможности и направления развития молекулярно-эпидемиологического мониторинга в надзоре за энтеровирусными инфекциями | |||
| Опыт Российской Федерации | |||
| Инфекция и иммунитет | |||
| Способ получения цианистых соединений | 1924 |
|
SU2018A1 |
| LU QB, et al | |||
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
Авторы
Даты
2021-12-16—Публикация
2020-09-15—Подача