Область техники
Настоящее изобретение относится к новой аденилосукцинат-синтетазе, к микроорганизму, содержащему ее, и к способу получения пуриновых нуклеотидов с использованием такого микроорганизма.
Предшествующий уровень техники
5'-Инозинмонофосфат (ниже IMP), вещество на основе нуклеиновых кислот, представляет собой промежуточное соединение биосинтетической метаболической системы нуклеиновых кислот, используемое в различных областях (например в лекарственных средствах, в различных медицинских применениях и т.д.), и является веществом, широко используемым в качестве пищевой приправы или в пище наряду с 5'-гуанинмонофосфатом (ниже GMP). Известно, что сам IMP создает вкус говядины и усиливает вкус мононатриевой соли глутаминовой кислоты (MSG) подобно GMP, таким образом привлекая общее внимание как вкусовая приправа на основе нуклеиновой кислоты.
Способы получения IMP могут включать способ ферментативного расщепления рибонуклеиновой кислоты, экстрагированной из дрожжевых клеток, способ химического фосфорилирования инозина, полученного ферментацией (Agri. Biol. Chem., 36, 1511(1972), etc.), способ культивирования микроорганизма, который непосредственно производит IMP и выделение IMP из культуральной среды и т.д. Среди этих способов наиболее широко используемым является использование микроорганизма, способного непосредственно продуцировать IMP.
Дополнительно способ получения GMP может включать способ превращения ксантозин-5'-монофосфата (ниже ХМР), продуцируемого посредством микробной ферментации, в GMP, с использованием коринеформных бактерий и способ превращения ХМР, продуцируемого посредством микробной ферментации, в GMP с использованием Escherichia coli. В вышеуказанных способах, когда GMP получают способом, в котором сначала продуцируют ХМР, а затем его превращают в GMP, производительность по ХМР (то есть предшественнику реакции превращения во время микробной ферментации) должна быть увеличена и, кроме того, и полученный ХМР и GMP, полученный уже в результате всего процесса реакции превращения, должны быть защищены от потерь.
При этом, поскольку ферменты в природе не всегда проявляют оптимальные свойства с точки зрения активности, стабильности, субстратной специфичности к оптическим изомерам и т.д., в промышленном применении были предприняты различные попытки улучшить ферменты для достижения необходимого использования путем изменения их аминокислотной последовательности. В их числе были использованы рациональное конструирование и сайт-направленный мутагенез ферментов для улучшения функций ферментов в некоторых случаях; однако, эти методы имеют недостатки, заключающиеся в том, что информация о структуре целевого фермента является недостаточной или не ясна взаимосвязь между структурой и функцией; таким образом, их нельзя эффективно использовать. В этом случае, как было сообщено, активность фермента может быть увеличена путем улучшения фермента с помощью метода направленной эволюции, в котором ферменты с желательными признаками отбирают из библиотеки мутантных ферментов, созданной посредством случайных вариаций генов фермента. Авторы настоящего изобретения провели обширные исследования для получения высокого выхода пуриновых нуклеотидов с помощью способа получения пуриновых нуклеотидов, содержащих IMP или ХМР, путем микробной ферментации. В результате они обнаружили белковые варианты, имеющие более высокое продуцирование пуриновых нуклеотидов, тем самым завершив настоящее изобретения.
Описание изобретения
Техническая задача
Задачей настоящего раскрытия является предложение варианта аденилосукцинат-синтетазы.
Другой задачей настоящего изобретения является предложение полинуклеотида, кодирующего вариант аденилосукцинат-синтетазы.
Еще одной задачей настоящего изобретения является предложение вектора, содержащего полинуклеотид.
Еще одной задачей настоящего изобретения является создание микроорганизма, способного продуцировать пуриновые нуклеотиды, который содержит вариант аденилосукцинат-синтетазы и вектор.
Еще одной задачей настоящего изобретения является создание способа получения пуриновых нуклеотидов, который включает культивирование микроорганизма рода Corynebacterium в среде; и выделение пуриновых нуклеотидов из микроорганизма или среды.
Техническое решение
Ниже типичные воплощения настоящего раскрытия будут описаны подробно. При этом, каждое из пояснений и типичных воплощений, раскрытых в данном документе, может быть применено к другим пояснениям и типичным воплощениям. То есть все комбинации различных факторов, раскрытых в данном документе, входят в объем настоящего раскрытия. Кроме того, объем настоящего раскрытия не следует ограничивать конкретным раскрытием, представленным ниже.
Для достижения вышеуказанных целей, в одном аспекте настоящего изобретения предложен вариант аденилосукцинат-синтетазы, в котором 85-я аминокислота с N-конца аминокислотной последовательности SEQ ID NO: 2 заменена другой аминокислотой. Модифицированная аденилосукцинат-синтетаза имеет модификацию аминокислоты в 85-м положении с N-конца аминокислотной последовательности SEQ ID NO: 2. В частности, в настоящем изобретении предложен вариант аденилосукцинат-синтетазы, имеющей по меньшей мере имеющей одно изменение в аминокислотной последовательности SEQ ID NO: 2, и эта модификация включает замену в 85-ом положении с N-конца другой аминокислотой.
При использовании здесь термин "аденилосукцинат-синтетаза" относится к ферменту, играющему важную роль в биосинтезе пурина. В контексте настоящего описания изобретения фермент относится к белку, участвующему в продуцировании пуриновых нуклеотидов, включая 5'-инозинмонофосфат (IMP) или 5'-ксантозинмонофосфат (ХМР).
В настоящем описании SEQ ID NO: 2 относится к аминокислотной последовательности, обладающей активностью аденилосукцинат-синтетазы. В частности, SEQ ID NO: 2 представляет собой последовательность белка, обладающего активностью аденилосукцинат-синтетазы, кодируемой геном purA. Аминокислотная последовательность SEQ ID NO: 2 может быть получена из NCBI GenBank, являющегося известной базой данных. В одном воплощении аминокислотная последовательность SEQ ID NO: 2 может происходить из микроорганизма рода Corynebacterium, без ограничения им, и может включать любую последовательность, обладающую такой же активностью, как указанная выше аминокислотная последовательность, без ограничения. Кроме того, в объем аминокислотной последовательности SEQ ID NO: 2 может входить аминокислотная последовательность SEQ ID NO: 2, обладающая активностью аденилосукцинат-синтетазы, или аминокислотная последовательность, имеющая 80% или более гомологии или идентичности с аминокислотой последовательностью SEQ ID NO: 2, без ограничения ими. В частности, вышеуказанная аминокислотная последовательность может включать аминокислотную последовательность SEQ ID NO: 2 и/или аминокислотную последовательность, имеющую по меньшей мере 80%, 85%, 90%, 95%, 96%, 97%, 98%, или 99%-ную или более гомологию или идентичность с аминокислотной последовательностью SEQ ID NO: 2. Аминокислотная последовательность, имеющая гомологию или идентичность, может представлять собой последовательность в вышеуказанном диапазоне, исключая последовательность, имеющую 100%-ную идентичность, или может быть последовательностью, имеющей идентичность менее чем 100%. Кроме того, очевидно, что любой белок, имеющий аминокислотную последовательность с делецией, модификацией, заменой или добавлением в части последовательности, также можно использовать в настоящем изобретении, если он имеет гомологию или идентичность и проявляет эффективность, соответствующую этому вышеуказанному белку.
В настоящем описании термин "вариант аденилосукцинат-синтетазы" может быть использован взаимозаменяемо с вариантом полипептида, обладающим способностью производить пуриновые нуклеотиды; пуриновые нуклеотиды-продуцирующим вариантом полипептида; вариантом полипептида, продуцирующим пуриновые нуклеотиды; вариантом полипептида, обладающим активностью аденилосукцинат-синтетазы; вариантом аденилосукцинат-синтетазы и т.д. Кроме того, белок может происходить из рода Corynebacterium stationis, но не ограничен им.
Вариант аденилосукцинат-синтетазы включает модификацию аминокислоты в 85-ом положении с N-конца в аминокислотной последовательности SEQ ID NO: 2. Вариант аденилосукцинат-синтетазы представляет собой вариант, в котором 85-я аминокислота в аминокислотной последовательности SEQ ID NO: 2 заменена другой аминокислотой. Вариант аденилосукцинат-синтетазы может включать аминокислотную последовательность SEQ ID NO: 2 или может представлять собой вариант аденилосукцинат-синтетазы, обладающий более слабой активностью по сравнению с аденилосукцинат-синтетазой, полученной из микроорганизма дикого типа. Такой вариант аденилосукцинат-синтетазы указывает на модификацию 85-й аминокислоты с N-конца в аминокислотной последовательности SEQ ID NO: 2 или аминокислотную последовательность, имеющую по меньшей мере 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99%-ную или более гомологию или идентичность с аминокислотной последовательности SEQ ID NO: 2, как объяснено выше.
В частности, вариант аденилосукцинат-синтетазы представляет собой вариант, в котором 85-я аминокислота в аминокислотной последовательности SEQ ID NO: 2 заменена серином, аланином, валином, лейцином, метионином, изолейцином, треонином, аспарагином, глутамином, цистеином, тирозином, лизином, аспарагиновой кислотой или глутаминовой кислотой, и вариант аденилосукцинат-синтетазы может иметь более слабую активность аденилосукцинат-синтетазы по сравнению с полипептидом, содержащим аминокислотную последовательность SEQ ID NO: 2, но вариант аденилосукцинат-синтетазы не ограничен ими.
В контексте настоящего изобретения, когда микроорганизм включает вариант аденилосукцинат-синтетазы, количество продуцируемых пуриновых нуклеотидов, включая IMP или ХМР, увеличивается. Это имеет значение, так как настоящее изобретение позволяет увеличивать количество продуцируемого IMP или ХМР с помощью варианта аденилосукцинат-синтетазы по настоящему изобретению, в то время как штамм Corynebacterium дикого типа не может продуцировать IMP или ХМР или может продуцировать только очень небольшое количество IMP или ХМР.
Вариант аденилосукцинат-синтетазы может содержать аминокислотную последовательность, выбранную из группы аминокислотных последовательностей, где 85-я аминокислота с N-конца в аминокислотной последовательности SEQ ID NO: 2 заменена аминокислотой, выбранной из группы состоящий из серина, аланина, валина, лейцина, метионина, изолейцина, треонина, аспарагина, глутамина, цистеина, тирозина, лизина, аспарагиновой кислоты и глутаминовой кислоты. В частности, вариант аденилосукцинат-синтетазы может состоять из полипептида, включающего аминокислотную последовательность, которая выбрана из группы аминокислотных последовательностей, где 85-я аминокислота с N-конца в аминокислотной последовательности SEQ ID NO: 2 заменена аминокислотой, выбранной из группы, состоящей из серина, аланина, валина, лейцина, метионина, изолейцина, треонина, аспарагина, глутамина, цистеина, тирозина, лизина, аспарагиновой кислоты и глутаминовой кислоты. Кроме того, вариант аденилосукцинат-синтетазы может включать аминокислотную последовательность, где 85-я аминокислота с N-конца в аминокислотной последовательности SEQ ID NO: 2 заменена другой аминокислотой, которая имеет аминокислотную последовательность варианта аденилосукцинат-синтетазы или аминокислотную последовательность, имеющую 80%-ную или более гомологию или идентичность аминокислотной последовательности варианта аденилосукцинат-синтетазы, но аминокислотная последовательность не ограничена ими. В частности, вариант аденилосукцинат-синтетазы по настоящему изобретению может включать полипептид, имеющий по меньшей мере 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99%-ную или более гомологию или идентичность с аминокислотной последовательностью, где 85-я аминокислота в аминокислотной последовательности SEQ ID NO: 2 заменена аминокислотой, выбранной из группы, состоящей из серина, аланина, валина, лейцина, метионина, изолейцина, треонина, аспарагина, глутамина, цистеина тирозин, лизин, аспарагиновой кислоты и глутаминовой кислот. Кроме того, очевидно, что любая аминокислотная последовательность, имеющая вышеуказанную гомологию или идентичность последовательности и проявляющая эффект, соответствующий эффекту белка, также должна входить в объем настоящего изобретения, даже если часть аминокислотной последовательности может иметь делецию, модификацию, замену или добавление в части последовательности, в дополнение к аминокислоте в 85-м положении.
То есть, хотя в настоящем описании изобретения описан "белок или полипептид, имеющий аминокислотную последовательность конкретной SEQ ID NO", очевидно, что белок, имеющий аминокислотную последовательность с делецией, модификацией, заменой или добавлением в части последовательности, также может быть использован в настоящем изобретении, если белок обладает активностью, идентичной или соответствующей активности полипептида, содержащего аминокислотную последовательность соответствующей SEQ ID NO. Например, пока белок имеет активность, идентичную или соответствующую активности варианта полипептида, не исключены добавления, природные мутации, молчащие мутации или консервативные замены, которые не изменяют функции белка, до или после аминокислотной последовательности. Очевидно, что белок, имеющий такое добавление или мутацию последовательности, также входит в объем настоящего изобретения.
"Консервативная замена" означает замену аминокислоты другой аминокислотой, имеющей похожие структурные и/или химические свойства. Такая аминокислотная замена обычно может происходить на основании сходства полярности остатков, заряда, растворимости, гидрофобности, гидрофильности и/или амфипатической природы. Например, положительно заряженные (основные) аминокислоты включают аргинин, лизин и гистидин; и отрицательно заряженные (кислотные) аминокислоты включают глутаминовую кислоту и аспарагиновую кислоту; ароматические аминокислоты включают фенилаланин, триптофан и тирозин; и гидрофобные аминокислоты включают аланин, валин, изолейцин, лейцин, метионин, фенилаланин, тирозин и триптофан.
Соответственно, в настоящем изобретении "вариант" может дополнительно включать консервативную замену и/или модификацию по меньшей мере одной аминокислоты в "белке или полипептиде, имеющем аминокислотную последовательность конкретной SEQ ID NO". Например, некоторые варианты могут включать варианты, в которых удалена по меньшей мере одна часть, такая как N-концевая лидерная последовательность или трансмембранный домен. Другие варианты могут включать варианты, в которых часть удалена с N-конца и/или С-конца зрелого белка. Вариант также может включать другие модификации, например делеции или добавления аминокислот, которые оказывают минимальное влияние на свойства и вторичную структуру полипептида. Например, полипептид может быть конъюгирован с сигнальной (или лидерной) последовательностью на N-конце белка, которая котрансляционно или пост-трансляционно направляет перенос белка. Полипептид также может быть конъюгирован с другой последовательностью или линкером для облегчения идентификации, очистки или синтеза полипептида. Термин "вариант" может быть использован взаимозаменяемо с модификацией, модифицированным белком, модифицированным полипептидом, мутантом, мутеином, дивергентом и т.д., и любой термин может использоваться без ограничения, если он используется в смысле "являющийся модифицированным".
Гомология и идентичность означают степень родства между двумя данными аминокислотными последовательностями или нуклеотидными последовательностями и могут быть выражены в процентах.
Термины "гомология" и "идентичность" часто могут использоваться взаимозаменяемо.
Гомология последовательности или идентичность консервативного полинуклеотида или полипептида может быть определена стандартным алгоритмом выравнивания, и штрафы за пробелы по умолчанию, установленные программой, которая будет использоваться, можно использовать в комбинации. По существу, гомологичные или идентичные последовательности могут гибридизироваться в умеренных или очень жестких условиях по всей длине их последовательности или по меньшей мере примерно на 50%, примерно на 60%, примерно на 70%, примерно на 80% или примерно на 90% всей длины. Что касается полинуклеотидов для гибридизации, также могут быть рассмотрены полинуклеотиды, содержащие вместо кодона вырожденный кодон.
Имеют ли какие-либо две полинуклеотидные или полипептидные последовательности гомологию, сходство или идентичность, можно определить, например, с помощью известного компьютерного алгоритма, такого как программа "FASTA" с использованием параметров по умолчанию, как в Pearson et al. (1988) (Proc. Natl. Acad. Sci. USA 85]: 2444). Альтернативно, они могут быть определены с использованием алгоритма Needleman-Wunsch (Needleman and Wunsch, 1970, J. Mol. Biol. 48: 443 to 453), как осуществлено в программе Needleman пакета EMBOSS (EMBOSS: The European Molecular Biology Open Software Suite, Rice et al, 2000, Trends Genet. 16: 276 - 277) (версия 5.0.0 или более поздняя) (включая пакет программ GCG (Devereux, J., et al, Nucleic Acids Research 12: 387 (1984)), BLASTP, BLASTN, FASTA (Atschul, [S.] [F.] et al., J Molec Biol 215]: 403 (1990); Guide to Huge Computers, Martin J. Bishop, [ED.,] Academic Press, San Diego, 1994, и [CARILLO ETA/.](1988) SIAM J Applied Math 48: 1073). Например, гомология, сходство или идентичность могут быть определены с использованием BLAST или ClustalW Национального центра биотехнологической информации.
Гомология, сходство или идентичность полинуклеотидов или полипептидов могут быть определены путем сравнения информации о последовательности с использованием компьютерной программы GAP (например Needleman et al. (1970), J Mol Biol 48: 443), как раскрыто в Smith and Waterman, Adv. Appl. Math (1981) 2:482. В кратком изложении, программа GAP определяет подобие как число выровненных символов (то есть нуклеотидов или аминокислот), которые являются подобными, деленное на общее количество символов в более короткой из двух последовательностей. Параметры по умолчанию для программы GAP могут включать: (1) унарную матрицу сравнения (содержащую значение 1 для идентичностей и 0 для не-идентичностей) и взвешенную матрицу сравнения (или EDNAFULL матрица замен (EMBOSS версия NCBI NUC4.4)) по Gribskov et al (1986) Nucl. Acids Res. 14: 6745, как описано в Schwartz and Dayhoff, eds., Atlas Of Protein Sequence And Structure, National Biomedical Research Foundation, pp.353-358 (1979); (2) штраф 3.0 за каждый пробел и дополнительный штраф 0.10 за каждый символ в каждом пробеле (или штраф за внесение пробела 10, штраф за продление пробела 0,5); и (3) отсутствие штрафа за конечные пробелы. Следовательно, термин "гомология" или "идентичность", используемый в данном документе, представляет соответствие последовательностей.
Кроме того, очевидно, что полинуклеотид, который может быть транслирован, из-за вырожденности кода, в вариант полипептида, содержащий аминокислотную последовательность, где 85-я аминокислота с N-конца аминокислотной последовательности SEQ ID NO: 2 заменена другой аминокислотой, или вариант полипептида, имеющий гомологию или идентичность с ним, также может быть включен. Кроме того, путем гибридизации в жестких условиях с зондом, который может быть получен из известной последовательности гена (например последовательности, комплементарной всей или части нуклеотидной последовательности), любая полинуклеотидная последовательность, кодирующая вариант аденилосукцинат-синтетазы, включая аминокислотную последовательность, где 85-я аминокислота аминокислотной последовательности SEQ ID NO: 2 заменена аминокислотой, выбранной из группы, состоящей из серина, аланина, валина, лейцина, метионина, изолейцина, треонина, аспарагина, глутамина, цистеина, тирозина, лизина, аспарагиновой кислоты и глутаминовой кислоты, может быть включена без ограничения.
Другой аспект настоящего изобретения относится к полинуклеотиду, кодирующему вариант аденилосукцинат-синтетазы или к вектору, включающему такой полинуклеотид.
При использовании здесь, термин "полинуклеотид" относится к цепи ДНК или РНК, имеющей более чем определенную длину, в виде нуклеотидного полимера, которая представляет собой длинную цепь нуклеотидных мономеров, соединенных ковалентными связями, и, более конкретно, к полинуклеотидному фрагменту, кодирующему вариант полипептида.
Полинуклеотид, кодирующий вариант полипептида по настоящему раскрытию, может включать любую полинуклеотидную последовательность без ограничения, при условии, что она кодирует вариант полипептида, обладающий активностью аденилосукцинат-синтетазы. В настоящем описании ген, кодирующий аминокислотную последовательность аденилосукцинат-синтетазы, представляет собой ген purA, и, в частности, ген может происходить из Corynebacterium stationis, без ограничения им.
В частности, из-за вырожденности кода или путем рассмотрения кодонов, являющихся предпочтительными для микроорганизма, в котором полипептид может экспрессироваться, в кодирующую область полинуклеотида могут быть внесены различные модификации, в объеме, который не изменяет аминокислотную последовательность полипептида. Любая полинуклеотидная последовательность может быть включена без ограничения, при условии, что она кодирует вариант аденилосукцинат-синтетазы, где 85-я аминокислота в аминокислотной последовательности SEQ ID NO: 2 заменена другой аминокислотой.
Кроме того, путем гибридизации в жестких условиях с зондом, который может быть получен из известной последовательности гена (например последовательности, комплементарной всей или части нуклеотидной последовательности), любая последовательность, кодирующая белок, обладающий активностью варианта аденилосукцинат-синтетазы, где 85-я аминокислота в аминокислотной последовательности SEQ ID NO: 2 заменена другой аминокислотой, может быть включена без ограничения.
"Жесткие условия" относятся к условиям, которые обеспечивают специфическую гибридизацию полинуклеотидов. Такие условия подробно описаны в литературе (например в J. Sambrook et al., выше). Жесткие условия могут включать условия, в которых гены, имеющие высокую гомологию или идентичность (например гены, имеющие 40% или более, в частности 90% или более, более конкретно 95% или более, еще более конкретно 97% или более, особенно конкретно 99%-ную или более гомологию или идентичность) могут гибридизироваться друг с другом; к условиям, в которых гены, имеющие более низкую гомологию или идентичность, не могут гибридизироваться друг с другом; или к условиями, которые являются обычными условиями промывки для Саузерн-гибридизации (например, концентрация соли и температура, соответствующие 60°С, 1 × SSC (раствор хлорида и цитрата натрия), 0,1% SDS (додецилсульфат натрия); в частности, 60°С, 0,1 × SSC, 0,1% SDS; более конкретно 68°С, 0,1 × SSC, 0,1% SDS, один раз, в частности, два или три раза).
Гибридизация требует, чтобы две нуклеиновые кислоты имели комплементарные последовательности, хотя, в зависимости от жесткости гибридизации, могут быть возможными несоответствия между основаниями. Термин "комплементарность" используется для описания взаимосвязи между нуклеотидными основаниями, которые могут гибридизироваться друг с другом. Например, в отношении ДНК, аденозин комплементарен тимину, а цитозин комплементарен гуанину. Соответственно, настоящее изобретение может также включать выделенные фрагменты нуклеиновых кислот, комплементарные всей последовательности, а также по существу тождественные последовательности нуклеиновых кислот.
В частности, полинуклеотид, имеющий гомологию или идентичность, может быть обнаружен с использованием условий гибридизации, включающих стадию гибридизации при Tm 55°С, и с использованием вышеуказанных условий. Кроме того, значение Tm может составлять 60°С, 63°С или 65°С, без ограничения им, и может соответствующим образом контролироваться специалистами в данной области техники в соответствии с поставленной целью.
Подходящая жесткость для гибридизации полинуклеотидов зависит от длины полинуклеотидов и степени комплементарности, и переменные хорошо известны в данной области (см. Sambrook et al., выше, от 9,50 до 9,51, от 11,7 до 11,8).
В настоящем описании ген, кодирующий аминокислотную последовательность варианта аденилосукцинат-синтетазы, представляет собой ген purA, и полинуклеотид, кодирующий ген, является таким, как пояснено выше.
В настоящем описании изобретения полинуклеотид, кодирующий вариант аденилосукцинат-синтетазы, также является таким, как пояснено выше.
При использовании здесь термин "вектор" относится к конструкции ДНК, содержащей нуклеотидную последовательность полинуклеотида, кодирующего полипептид-мишень, которая функционально связана с подходящей контрольной последовательностью, так что целевой полипептид экспрессируется в подходящем хозяине. Контрольная последовательность может включать промотор для инициации транскрипции, любую последовательность оператора для контроля такой транскрипции, последовательность, кодирующую соответствующий сайт связывания рибосомы на мРНК, и последовательность контроля терминации транскрипции и трансляции. После трансформации в подходящего хозяина, вектор может реплицироваться или функционировать независимо от генома хозяина или может интегрироваться в сам геном.
Вектор, используемый в настоящем изобретении, может не быть конкретно ограничен, если этот вектор реплицируется в клетке-хозяине, и можно использовать любой вектор, известный в данной области техники. Примеры обычно используемого вектора могут включать природные или рекомбинантные плазмиды, космиды, вирусы и бактериофаги. Например, в качестве фагового вектора или космидного вектора можно использовать pWE15, М13, MBL3, MBL4, IXII, ASHII, APII, t10, t11, Charon4A, Charon21A, и т.д. и в качестве плазмидного вектора - основанные на pBR, pUC, pBluescriptII, pGEM, pTZ, pCL, pET, и т.д. В частности, можно использовать вектор pDZ, pACYC177, pACYC184, pCL, pECCG117, pUC19, pBR322, pMW118, pCC1BAC и т.д.
В одном воплощении полинуклеотид, кодирующий целевой полипептид, может быть встроен в хромосому с помощью вектора для хромосомной вставки. Вставка полинуклеотида в хромосому может быть осуществлена с использованием любого способа, известного в данной области (например посредством гомологичной рекомбинации), но способ не ограничен ими. Может быть дополнительно включен селективный маркер для подтверждения вставки вектора в хромосому. Селективный маркер использовали для отбора клеток, трансформированных вектором (то есть для подтверждения присутствия вставки целевой молекулы нуклеиновой кислоты), а также могут быть использованы маркеры, способные обеспечивать селективные фенотипы (например лекарственную резистентность, ауксотрофию, резистентность к цитотоксическим агентам и экспрессию поверхностных полипептидов). В условиях обработки селективными агентами, только клетки, способные экспрессировать маркеры селекции, могут выжить или экспрессировать другие фенотипические признаки, и, таким образом, трансформированные клетки могут быть легко отобраны.
Еще один аспект настоящего изобретения относится к микроорганизму, продуцирующему пуриновые нуклеотиды благодаря тому, что он содержит вариант аденилосукцинат-синтетазы или полинуклеотид, кодирующий вариант аденилосукцинат-синтетазы. В частности, микроорганизм, содержащий вариант аденилосукцинат-синтетазы и/или полинуклеотид, кодирующий вариант аденилосукцинат-синтетазы, может представлять собой микроорганизм, полученный посредством трансформации с использованием вектора, содержащего полинуклеотид, но микроорганизм не ограничен этим.
При использовании здесь термин "трансформация" относится к процессу введения вектора, который включает полинуклеотид, кодирующий целевой белок, в клетку-хозяина, так что белок, кодируемый этим полинуклеотидом, может экспрессироваться в клетке-хозяине. Не имеет значения, встроен ли трансформированный полинуклеотид в хромосому клетки-хозяина и расположен на ней или расположен вне хромосомы, при условии, что трансформированный полинуклеотид может экспрессироваться в клетке-хозяине. Кроме того, полинуклеотид может включать ДНК и РНК, кодирующие целевой белок. Полинуклеотид может быть введен в любой форме, при условии, что этот полинуклеотид может быть введен в клетку-хозяина и экспрессироваться в ней. Например, полинуклеотид может быть введен в клетку-хозяина в форме экспрессионной кассеты, которая представляет собой генную конструкцию, включающую все элементы, необходимые для ее автономной экспрессии. Экспрессионная кассета может включать промотор, сигнал терминации транскрипции, сайт связывания рибосомы и сигнал терминации трансляции, которые могут быть функционально связаны с полинуклеотидом. Экспрессионная кассета может находиться в форме вектора экспрессии, осуществляющего саморепликацию. Кроме того, полинуклеотид может быть введен в клетку-хозяина, так чтобы быть функционально связанным с последовательностью, необходимой для экспрессии в клетке-хозяине, без ограничения этим.
Кроме того, термин "функционально связан" относится к функциональной связи между последовательностью гена и последовательностью промотора, которая инициирует и опосредует транскрипцию полинуклеотида, кодирующего целевой полипептид по настоящему изобретению.
При использовании здесь термин "микроорганизм, включающий вариант полипептида" или "микроорганизм, включающий вариант аденилосукцинат-синтетазы", относится к микроорганизму, обеспечивающему продуцирование IMP или продуцирование ХМР в микроорганизме, который в природных условиях имеет слабое продуцирование IMP, или его родительский штамм не имеет продуцирования IMP или продуцирования ХМР. В частности, микроорганизм может представлять собой микроорганизм, экспрессирующий вариант аденилосукцинат-синтетазы, включающий по меньшей мере одну аминокислотную замену в аминокислотной последовательности SEQ ID NO: 2, и модификация аминокислоты может включать замену 85-й аминокислоты с N-конца аминокислотной последовательности SEQ ID NO: 2 другой аминокислотой. Кроме того, микроорганизм может представлять собой микроорганизм, который экспрессирует вариант полипептида, обладающий активностью аденилосукцинат-синтетазы, где 85-я аминокислота в аминокислотной последовательности SEQ ID NO: 2 заменена другой аминокислотой, но микроорганизм не ограничен этим.
Микроорганизм может представлять собой клетку или микроорганизм, который содержит полинуклеотид, кодирующий вариант аденилосукцинат-синтетазы, или клетку или микроорганизм, который трансформирован вектором и способен экспрессировать вариант аденилосукцинат-синтетазы. В контексте настоящего изобретения клетка-хозяин или микроорганизм может представлять собой любой микроорганизм, который может экспрессировать пуриновые нуклеотиды благодаря содержащемуся варианту аденилосукцинат-синтетазы.
В настоящем описании изобретения термин "микроорганизм, продуцирующий пуриновые нуклеотиды" может быть использован взаимозаменяемо с "пуриновый нуклеотид-продуцирующим микроорганизмом" и "микроорганизмом, продуцирующим пуриновые нуклеотиды".
В контексте настоящего описания термин "пуриновый нуклеотид" относится к нуклеотиду содержащему структуру на основе пурина, например IMP или ХМР, но пуриновый нуклеотид не ограничен ими.
В настоящем раскрытии термин "микроорганизм, продуцирующий пуриновые нуклеотиды" может представлять собой микроорганизм, у которого произошла генетическая модификация или была повышена активность для продуцирования желательных пуриновых нуклеотидов, включая как микроорганизм дикого типа, так и микроорганизмы с естественной или искусственной генетической модификацией, и микроорганизм может представлять собой микроорганизм, в котором определенный механизм усилен или ослаблен по таким причинам, как вставка экзогенного гена, усиление или инактивация активности эндогенного гена и т.д. В контексте настоящего изобретения микроорганизм, продуцирующий пуриновые нуклеотиды, характеризуется тем, что он имеет повышенное продуцирование желательных пуриновых нуклеотидов за счет содержащегося варианта аденилосукцинат-синтетазы и, в частности, микроорганизм может представлять собой микроорганизм рода Corynebacterium. В частности, микроорганизм, продуцирующий пуриновые нуклеотиды, или микроорганизм, обладающий способностью продуцировать пуриновые нуклеотиды, может представлять собой микроорганизм, в котором часть гена, участвующего в пути биосинтеза пуриновых нуклеотидов, усилена или ослаблена, или часть гена, участвующего в пути деградации пуриновых нуклеотидов, усилена или ослаблена. Например, микроорганизм может представлять собой микроорганизм, в котором экспрессия purF, кодирующего фосфорибозилпирофосфат-амидотрансферазу, усилена или экспрессия guaB, кодирующего инозин-5'-монофосфатдегидрогеназу, соответствующую пути деградации IMP, ослаблена, но микроорганизм не ограничен ими.
При использовании здесь, термин "микроорганизм рода Corynebacterium, продуцирующий 5'-пуриновые нуклеотиды" относится к микроорганизму рода Corynebacterium, который продуцирует пуриновые нуклеотиды естественным образом или в результате модификации. В частности, при использовании здесь, микроорганизм рода Corynebacterium, продуцирующий пуриновые нуклеотиды, может представлять собой микроорганизм рода Corynebacterium, который имеет улучшенное продуцирование пуриновых нуклеотидов за счет усиления или ослабления активности гена purA, кодирующего аденилосукцинат-синтетазу. Более конкретно, при использовании в данном описании, микроорганизм рода Corynebacterium, продуцирующий пуриновые нуклеотиды, может представлять собой микроорганизм рода Corynebacterium, который обладает улучшенным продуцированием пуриновых нуклеотидов за счет включения варианта аденилосукцинат-синтетазы по настоящему изобретению или полинуклеотида, кодирующего ее, или будучи трансформированным вектором, включающим полинуклеотид, кодирующий вариант аденилосукцинат-синтетазы. "Микроорганизм рода Corynebacterium, имеющий улучшенное продуцирование пуриновых нуклеотидов" относится к микроорганизму, имеющему повышенное продуцирование пуриновых нуклеотидов по сравнению с его родительским штаммом до трансформации или нонвариантным микроорганизмом. "Нонвариантный микроорганизм" относится к самому штамму дикого типа, микроорганизму, который не содержит вариант белка, продуцирующий пуриновые нуклеотиды, или к микроорганизму, который не трансформирован вектором, содержащим полинуклеотид, кодирующий вариант аденилосукцинат-синтетазы.
При использовании здесь "микроорганизм рода Corynebacterium" может представлять собой, в частности, Corynebacterium glutamicum, Corynebacterium ammoniagenes, Brevibacterium lactofermentum, Brevibacterium flavum, Corynebacterium thermoaminogenes, Corynebacterium efficiens, Corynebacterium stationis, и т.д., но микроорганизм не ограничен ими.
В еще одном аспекте настоящего изобретения предложен способ получения пуриновых нуклеотидов, включающий культивирование микроорганизма рода Corynebacterium, который продуцирует пуриновый нуклеотид, в среде, и извлечение пуриновых нуклеотидов из микроорганизма или среды.
В вышеуказанном способе культивирование микроорганизма может быть выполнено при помощи известной периодической культуры, непрерывной культуры, культуры с подпиткой и т.д., но способ культивирования конкретно не ограничен ими. В частности, условия культивирования могут не быть конкретно ограничены, но оптимальное значение рН (например рН 5-9, конкретно рН 6-8, и наиболее конкретно 6,8) может быть отрегулировано с использованием щелочного соединения (например гидроксида натрия, гидроксида калия или аммиака) или кислого соединения (например фосфорной кислоты или серной кислоты), а аэробные условия могут поддерживаться путем добавления кислорода или кислородсодержащей газовой смеси в культуру. Температуру культивирования можно поддерживать на уровне от 20 до 45°С, в частности от 25 до 40°С, и культивирование можно осуществлять в течение от примерно 10 часов до примерно 160 часов, но условия этим не ограничены. 5'-Инозиновая кислота, полученная в результате культивирования, может секретироваться в среду или может оставаться в клетках.
Кроме того, в культуральной среде, используемой в качестве источника углерода, сахара и углеводы (например глюкоза, сахароза, лактоза, фруктоза, мальтоза, патока, крахмал и целлюлоза), масла и жиры (например соевое масло, масло семян подсолнечника, арахисовое масло и кокосовое масло), жирные кислоты (например пальмитиновая кислота, стеариновая кислота и линолевая кислота), спирты (например глицерин и этанол), органические кислоты (например уксусная кислота) и т.д. могут быть использованы по отдельности или в комбинации, но источник углерода не ограничен ими. В качестве источника азота можно использовать азотсодержащие органические соединения (например пептон, дрожжевой экстракт, мясной экстракт, солодовый экстракт, кукурузный экстракт, соевую муку и мочевину) или неорганические соединения (например сульфат аммония, хлорид аммония, фосфат аммония, карбонат аммония и нитрат аммония) и т.д. отдельно или в комбинации, но источник азота не ограничен ими. В качестве источника фосфора можно использовать дигидрофосфат калия, гидрофосфат калия, соответствующую натрий-содержащую соль и т.д., но источник фосфора не ограничен ими. Кроме того, среда может также включать необходимые вещества, способствующие росту, такие как соли других металлов (например сульфат магния или сульфат железа), аминокислоты и витамины.
Способ извлечения пуриновых нуклеотидов, полученных на стадии культивирования по настоящему изобретению, заключается в сборе нужных пуриновых нуклеотидов из культуры с использованием подходящего способа, известного в данной области техники, в соответствии со способом культивирования. Например, можно использовать центрифугирование, фильтрацию, анионообменную хроматографию, кристаллизацию, ВЭЖХ и т.д., и нужные пуриновые нуклеотиды могут быть извлечены из среды или микроорганизма с использованием подходящего способа, известного в данной области.
Кроме того, стадия выделения может включать процесс очистки. Процесс очистки может быть выполнен с использованием подходящего способа, известного в данной области. Следовательно, выделенные пуриновые нуклеотиды могут находиться в очищенной форме или в микробной ферментационной жидкости, содержащей пуриновые нуклеотиды (Introduction to Biotechnology and Genetic Engineering, A. J. Nair, 2008).
Полезные эффекты изобретения
Когда микроорганизм рода Corynebacterium продуцирует пуриновые нуклеотиды с использованием варианта аденилосукцинат-синтетазы по настоящему изобретению, можно получать пуриновые нуклеотиды с высоким выходом.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Ниже настоящее изобретение будет подробно описано посредством типичных воплощений. Однако специалисту в области техники, к которой относится настоящее раскрытие, очевидно, что эти типичные воплощения предоставлены только для иллюстрации и не предназначены ограничивать объем настоящего изобретения.
Пример 1: Получение IMP-продуцирующего штамма на основе дикого типа
Штамм дикого типа рода Corynebacterium не может продуцировать IMP совсем или может продуцировать лишь очень незначительное количество, даже если это возможно. Соответственно, ГМР-продуцирующий штамм был получен на основе Corynebacterium stationis АТСС6872. Более конкретно, ГМР-продуцирующий штамм был получен путем усиления активности гена purF, кодирующего фосфорибозилпирофосфат-амидотрансферазу, которая является первым ферментом в биосинтезе пурина, и ослабления активности гена guaB, кодирующего дегидрогеназу 5'-инозиновой кислоты, которая соответствует пути деградации IMP.
Пример 1-1: Получение purF-усиленного штамма
Для получения штамма, в котором стартовый кодон гена purF модифицирован, получали инсерционный вектор, содержащий ген purF с SEQ ID NO: 3. Для клонирования гена purF в инсерционный вектор, в частности, проводили ПЦР с использованием геномной ДНК Corynebacterium stationis АТСС6872 в качестве матрицы и праймеров с SEQ ID NO: 4 и 5 и SEQ ID NO: 6 и 7 в течение 30 циклов денатурации при 94°С длительностью 30 с, отжига при 55°С в течение 30 с и удлинения при 72°С в течение 2 мин. ПЦР снова проводили с использованием двух фрагментов ДНК, полученных с помощью вышеуказанной ПЦР в качестве матрицы и праймеров SEQ ID NO: 4 и 72 в течение 30 циклов денатурации при 94°С длительностью 30 с, отжига при 55°С в течение 30 с и удлинения при 72°С в течение 2 мин с получением фрагментов ДНК. Полученные фрагменты ДНК расщепляли рестриктазой XbaI и клонировали в вектор pDZ (патент Кореи 10-0924065 и международная публикация 2008-033001), расщепленный тем же ферментом. Полученный таким образом вектор был назван pDZ-purF-g1a.

Рекомбинантный вектор pDZ-purF-g1a был трансформирован в Corynebacterium stationis АТСС6872 посредством электропорации, и штаммы, в геномную ДНК которых был встроен вектор путем гомологичной рекомбинации, были отобраны на среде, содержащей 25 мг/л канамицина. Выбранные первичные штаммы подвергали вторичному кроссинговеру, и эти отобранные штаммы подвергали секвенированию, посредством чего был выбран нужный штамм, в который была введена мутация. Штамм был назван штаммом ATCC6872::purF (g1a).
Пример 1-2: Получение guaB-ослабленного штамма
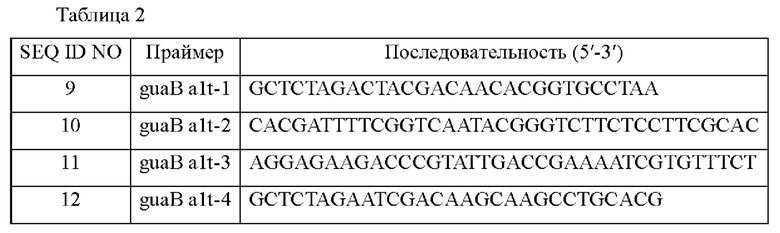
Для получения штамма, в котором модифицирован стартовый кодон гена guaB, получали инсерционный вектор, содержащий ген guaB с SEQ ID NO: 8. Для клонирования гена guaB в инсерционный вектор, в частности, проводили ПЦР с использованием геномной ДНК Corynebacterium stationis АТСС6872 в качестве матрицы и праймеров SEQ ID NO: 9 и 10 и SEQ ID NO: 11 и 12. Продукты ПЦР клонировали, как в Примере 1-1, и полученный вектор был назван pDZ-guaB-a1t. Этот вектор вводили в ATCC6872::purF (g1a), и был окончательно выбран штамм, в который была введена указанная выше мутация.
Окончательно выбранный штамм, продуцирующий IMP, на основе штамма дикого типа Corynebacterium stationis АТСС6872, был назван CJI2330.
Пример 1-3: Ферментативный титриметрический анализ CJI2330
После распределения посевной культуральной среды (2 мл) в пробирки (диаметр: 18 мм), пробирки автоклавировали. Каждый из АТСС6872 и CJI2330 инокулировали и инкубировали при 30°С в течение 24 ч при встряхивании и использовали в качестве посевной культуры. Ферментационную среду (29 мл) помещали в каждую встряхиваемую колбу Эрленмейера объемом 250 мл и автоклавировали при 121°С в течение 15 мин. Посевную культуру (2 мл) инокулировали в среду и культивировали в течение 3 суток. Условия культивирования соответствовали 170 об/мин, 30°С и рН 7,5.
После завершения культивирования количество продуцируемого IMP измеряли с помощью ВЭЖХ (SHIMAZDU LC20A), и результаты культивирования были такие, как представлено в таблице 3 ниже. Следующие результаты показывают, что штамм с усиленным purF и ослабленным purA продуцирует IMP.

- Посевная культуральная среда: 1% глюкозы, 1% пептона, 1% мясного экстракта, 1% дрожжевого экстракта, 0,25% хлорида натрия, 100 мг/л аденина, 100 мг/л гуанина, рН 7,5;
- Ферментационная среда: 0,1% глутамата натрия, 1% хлорида аммония, 1,2% сульфата магния, 0,01% хлорида кальция, 20 мг/л сульфата железа, 20 мг/л сульфата марганца, 20 мг/л сульфата цинка, 5 мг/л сульфата меди, 23 мг/л L-цистеина, 24 мг/л аланина, 8 мг/л никотиновой кислоты, 45 мкг/л биотина, 5 мг/л тиамина гидрохлорида, 30 мг/л аденина, 1,9% фосфорной кислоты (85%), 2,55% глюкозы, 1,45% фруктозы
Пример 2: Получение ослабленного варианта аденилосукцинат-синтетазы
Чтобы найти вариант аденилосукцинат-синтетазы, способный улучшить
продуцирование пуриновых нуклеотидов, получали библиотеку мутантов гена purA,
кодирующего аденилосукцинат-синтетазу.
Пример 2-1: Получение вектора, содержащего ген purA
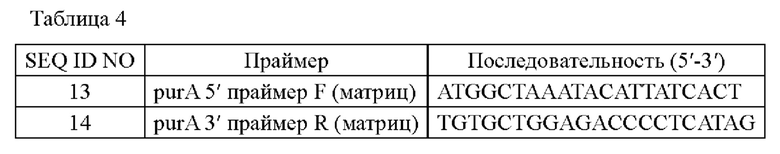
Для получения библиотеки мутантов гена purA сначала получали рекомбинантный вектор, содержащий ген purA. ПЦР проводили с использованием геномной ДНК Corynebacterium stationis АТСС6872 в качестве матрицы и праймеров SEQ ID NO: 13 и SEQ ID NO: 14, и продукт ПЦР клонировали в вектор pCR2.1 E.coli с использованием набора для клонирования ТОРО (Invitrogen) с получением pCR-guaB.
Пример 2-2: Получение библиотеки мутантов гена purA
Библиотеку мутантов гена purA получали на основании вектора, полученного в Примере 2-1. Библиотеку получали с использованием набора для допускающей ошибки ПЦР (Clontech Diversify® PCR Random Mutagenesis Kit). В условиях, в которых могли произойти мутации, проводили ПЦР с использованием праймеров SEQ ID NO: 15 и SEQ ID NO: 16. В частности, в условиях, когда могут происходить от 0 до 3 мутаций на 1000 п. н., предварительный нагрев проводили при 94°С в течение 30 с, а затем 25 циклов при 94°С в течение 30 с и 68°С в течение 1 мин 30 с. Полученный таким образом ПЦР-продукт подвергали ПЦР с использованием мегапраймера (от 500 нг до 125 нг) в течение 25 циклов при 95°С в течение 50 с, 60°С в течение 50 с и 68°С в течение 12 мин, обрабатывали DpnI и трансформировали в E.coli DH5a и распределяли на твердой среде LB, содержащей канамицин (25 мг/л). После отбора 20 разных видов трансформированных колоний, из них получали плазмиды и подвергали анализу секвенирования. В результате было подтверждено, что мутации были введены в разные сайты с частотой 2 мутации/кб. Собирали примерно 20000 трансформированных колоний Е. coli, экстрагировали плазмиды и назвали библиотекой pTOPO-purA.

Пример 3: Оценка полученной библиотеки и выбор штамма
Пример 3-1: Оценка библиотеки
Библиотеку рТОРО-purA, полученную в Примере 2-2, трансформировали в штамм CJI2330, полученный в Примере 1, посредством электропорации, и штамм был распределяли по питательной среде, содержащей 25 мг/л канамицина, с получением 10000 колоний, в которые был введен мутантный ген. Каждая из колоний была названа от CJI2330::pTOPO_purA (mt) 1 до CJI2330::pTOPO_purA (mt) 10000.
- Питательная среда: 1% пептона, 1% мясного экстракта, 0,25% хлорида натрия, 1% дрожжевого экстракта, 2% агара, рН 7,2.
Каждую из полученных 10000 колоний инокулировали в 200 мкл автоклавированной посевной культуральной среды и культивировали в 96-луночном планшете при встряхивании при 30°С, 1200 об/мин в течение 24 часов с использованием шейкера для микропланшетов (TAITEC) и использовали в качестве посевной культуры. Автоклавированную ферментационную среду (290 мкл) помещали в 96-луночный планшет и в нее инокулировали 20 мкл каждой из посевных культур, затем культивировали со встряхиванием в тех же условиях, что и выше, в течение 72 часов.
Для анализа 5'-инозиновой кислоты, продуцируемой в культуральной среде, после завершения культивирования, 3 мкл культурального супернатанта переносили в 96-луночный УФ-планшет, где каждая лунка содержала 197 мкл дистиллированной воды, и встряхивали в течение 30 секунд, используя считывающее устройство для микропланшетов, и измеряли оптическую плотность при 270 нм при 25°С с использованием спектрофотометра. Поглощение сравнивали с поглощением штамма CJI2330, и выбирали 50 колоний мутантных штаммов, демонстрирующих увеличение поглощения на 10% или более. Другие колонии демонстрировали похожее или пониженное поглощением по сравнению с контролем.
Поглощение 50 выбранных штаммов измеряли таким же образом, как выше, чтобы повторно исследовать количество продуцируемой 5'-инозиновой кислоты. Был выбран один штамм, CJI2330::pTOPO_purA (mt) 333, который показал значительное улучшение продуцирования 5'-инозиновой кислоты по сравнению со штаммом CJI2330.
Для подтверждения достоверности выбранных мутантов был проведен ферментативный титриметрический анализ.
После распределения посевной культуральной среды (2 мл) в аналитические пробирки (диаметр: 18 мм), эти пробирки автоклавировали. Каждый из CJI2330 и CJI2330::pTOPO_purA (mt) 333 инокулировали и инкубировали при 30°С в течение 24 часов при встряхивании и использовали в качестве посевной культуры. Ферментационную среду (29 мл) помещали в каждую встряхиваемую колбу Эрленмейера объемом 250 мл и автоклавировали при 121°С в течение 15 минут. Посевную культуру (2 мл) инокулировали в среду и культивировали в течение 3 суток. Условия культивирования соответствовали 170 об/мин, 30°С и рН 7,5.
После завершения культивирования количество продуцируемого IMP измеряли с помощью ВЭЖХ (SHIMADZU LC20A), и результаты культивирования были такими, как представлено в таблице 6 ниже.

Как видно из приведенных выше результатов, было подтверждено, что количество IMP увеличивалось примерно на 122% в штамме, где вектор содержал мутацию гена purA по сравнению со штаммом CJI2330. Соответственно, было установлено, что выбранная мутация в библиотеке была эффективной.
Пример3-2: Подтверждение изменения гена purA
Для подтверждения изменения генов мутантного штамма выполняли ПЦР в штамме CJI2330::pTOPO_purA(mt)333 с использованием праймеров SEQ ID NO: 17 и 18, и продукт ПЦР подвергали секвенированию, подтверждая тем самым наличие изменений в гене purA.

В частности, было подтверждено, что ген purA штамма СЛ2330::рТОРО_purA (mt) 333 включает вариант, где 85-я аминокислота (т.е. глицин) аминокислотной последовательности purА, представленной SEQ ID NO: 2, заменена серином (т.е. 253-й нуклеотид "g" заменен нуклеотидом "а"). Соответственно, в Примерах, приведенных ниже, были предприняты попытки подтвердить, может ли вышеуказанное изменение влиять на количество продуцируемых пуриновых нуклеотидов в каждом микроорганизме рода Corynebacterium.
Пример 4: Подтверждение продуцирования IMP в IMP-продуцирующем штамме, происходящем от АТСС6872
Был получен штамм-продуцент IMP из АТСС6872 и изменение, подтвержденное в Примере 3, было введено в этот штамм, и было подтверждено продуцирование IMP штаммом.
Пример 4-1: Выбор IMP-продуцирующего штамма, происходящего от АТСС6872
Для получения штамма-продуцента IMP, происходящего от штамма АТСС6872, культуру АТСС6872 суспендировали в фосфатном буфере (рН 7,0) или цитратном буфере (рН 5,5) с плотностью от 107 до 108 клеток/мл и обрабатывали ультрафиолетовым излучением при комнатной температуре или при 32°С в течение от 20 до 40 мин, чтобы индуцировать мутацию. Штамм дважды промывали 0,85% солевым раствором и после разбавления распределяли по минимальной среде, содержащей 1,7% агара, с добавлением в подходящей концентрации вещества, обеспечивающего резистентность, и таким образом получали колонии. Каждую колонию культивировали в питательной среде и затем культивировали в посевной культуральной среде в течение 24 часов. После культивирования каждой колонии в ферментационной среде в течение 3-4 суток, отбирали колонии, которые демонстрировали превосходное продуцирование IMP, накапливаемого в культуральной среде. Для получения штамма, продуцирующего IMP в высокой концентрации, обеспечивали аденин-ауксотрофный, гуанин-ликовый тип, чувствительность к лизоциму, резистентность к 3,4-дегидропролину, резистентность к стрептомицину, резистентность к сульфагуанидину, резистентность к норвалину и резистентность к триметоприму путем выполнения соответствующих процедур последовательным образом. В результате был окончательно выбран штамм CJI2335, обладающий резистентностью к вышеуказанным веществам и имеющий превосходное продуцирование IMP. Сравнивали резистентности штамма CJI2332 относительно резистентностей АТСС6872 и результаты представлены в следующей ниже Таблице 8.

- Минимальная среда: 2% глюкозы, 0,3% сульфата натрия, 0,1% монокалия фосфата, 0,3% дикалий фосфата, 0,3% сульфата магния, 10 мг/л хлорида кальция, 10 мг/л сульфата железа, 1 мг/л сульфата цинка, 3,6 мг/л хлорида марганца, 20 мг/л L-цистеина, 10 мг/л пантотената кальция, 5 мг/л тиамина гидрохлорида, 30 мкг/л биотина, 20 мг/л аденина, 20 мг/л гуанина, доведенная до рН 7,3.
Пример 4-2: Ферментативный титриметрический анализ CJI2332
После распределения среды для посева (2 мл) в аналитические пробирки (диаметр: 18 мм), эти пробирки автоклавировали. Каждый из АТСС6872 и CJI2332 инокулировали и инкубировали при 30°С в течение 24 часов при встряхивании и использовали в качестве посевной культуры. Ферментационную среду (29 мл) помещали в каждую встряхиваемую колбу Эрленмейера объемом 250 мл и автоклавировали при 121°С в течение 15 мин. Посевную культуру (2 мл) инокулировали в среду и культивировали в течение 3 суток. Условия культивирования соответствовали 170 об/мин, 30°С и рН 7,5.
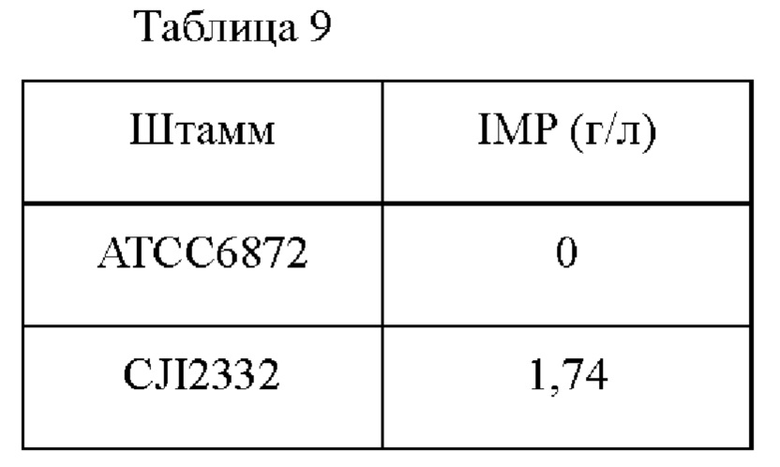
После завершения культивирования количество продуцированного IMP измеряли с помощью ВЭЖХ (SHIMADZU LC20A), и результаты культивирования представлены в Таблице 9 ниже.
Пример 4-3: Получение инсерционного вектора, содержащего вариант purA
Чтобы ввести варианты, выбранные в примере 3, в штаммы, получали инсерционный вектор. Процесс получения вектора для введения варианта purA (G85S) представляет собой следующее. Выполняли ПЦР с использованием CJI2330::Topo_purA (G85S) в качестве матрицы и праймеров SEQ ID NO: 19 и SEQ ID NO: 20. ПЦР проводили следующим образом: денатурация при 94°С в течение 5 мин; 20 циклов денатурации при 94°С в течение 30 с, отжиг при 55°С в течение 30 с и полимеризация при 72°С в течение 1 мин; и полимеризация при 72°С в течение 5 мин. каждый из полученных таким образом фрагментов генов переваривали с помощью XbaI. Каждый фрагмент гена клонировали в линейный вектор pDZ, переваренный XbaI, с использованием лигазы Т4, и таким образом был получен вектор pDZ-purA(G85S).
Пример 4-4: Введение вариантов в штаммы CJI2330 и CJI2332, полученные из АТСС6872, и их оценка
Варианты purA вводили в каждый из происходящих от дикого типа IMP-продуцирующих штаммов CJI2330, полученных в Примере 1, и штамм CJI2332, выбранный в Примере 4-1, и оценивали количество IMP, продуцируемое каждым штаммом. Чтобы подтвердить наличие изменения в гене purA, хромосомную ДНК штамма CJI2332 амплифицировали с помощью ПЦР. В частности, сначала фрагменты гена purA амплифицировали с помощью ПЦР с использованием хромосомной ДНК штамма CJI2332 в качестве матрицы и праймеров SEQ ID NO: 17 и 18, где ПЦР проводили с 28 циклами денатурации при 94°С в течение 1 мин; отжига при 58°С в течение 30 с и полимеризации при 72°С в течение 2 мин с использованием ДНК-полимеразы Taq. Нуклеотидные последовательности амплифицированных фрагментов purA анализировали с использованием тех же праймеров, и в результате было подтверждено, что в гене purA штамма CJI2332 изменений не было.
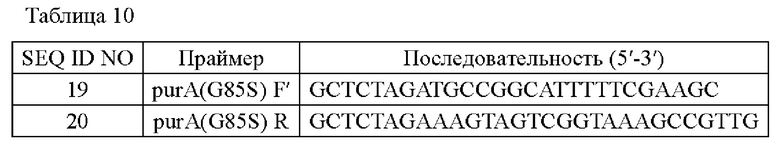
Затем вектор pDZ-purA (G85S) трансформировали в штамм CJI2330 и штамм CJI2332, и штаммы, где вектор был введен в геномную ДНК посредством рекомбинации гомологичных последовательностей, отбирали на среде, содержащей канамицин (25 мг/л). Отобранные первичные штаммы подвергали вторичному кроссинговеру, и, таким образом, отбирали штаммы, в которые был введен вариант целевого гена. Для подтверждения введения варианта гена в нужные трансформированные штаммы, выполняли ПЦР с использованием праймеров SEQ ID NO: 17 и SEQ ID NO: 18, и продукты ПЦР подвергали секвенированию. В результате было подтверждено, что варианты генов были введено в штаммы. Полученные таким образом штаммы были названы CJI2330::purA (G85S) и CJI2332::purA (G85S) соответственно.
Оценивали продуцирование IMP для каждого штамма CJI2330, CJI2332, CJI2330::purA (G85S) и CJI2332::purA (G85S). После завершения культивирования количество IMP, продуцируемого каждым штаммом, измеряли методом с использованием ВЭЖХ, и результаты культивирования показаны в таблице 11 ниже.

Приведенные выше результаты подтверждают, что штамм, в который был введен вариант гена purA, показал увеличение продуцирования IMP на 122% и примерно 116% по сравнению происходящих от дикого типа IMP-продуцирующими штаммами CJI2330 и CJI2332 соответственно.
Штамм CJI2332 был депонирован в Корейском центре культур микроорганизмов (KCCM) 22 июня 2018 года в соответствии с положениями Будапештского договора и ему присвоен регистрационный номер KCCM12277P. Кроме того, полученный штамм CJI2332::purA (G85S), также называемый CJI2348, был депонирован в KCCM 22 июня 2018 года в соответствии с положениями Будапештского договора и ему присвоен регистрационный номер KCCM12280P.
Пример 5: Подтверждение продуцирования 5'-ксантиловой кислоты с помощью варианта гена purA
Пример 5-1: Отбор ХМР-продуцирующих штаммов, происходящих от АТСС6872
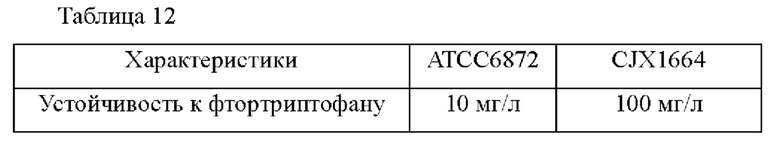
Для получения штамма, продуцирующего 5'-ксантозинмонофосфат (ХМР), происходящего от АТСС6872, штамм Corynebacterium stationis АТСС6872 суспендировали в фосфатном (рН 7,0) или цитратном буфере (рН 5,5) с плотностью от 107 клеток/мл до 108 клеток/мл и обрабатывали ультрафиолетовым излучением при комнатной температуре или при 32°С в течение от 20 минут до 40 минут, чтобы индуцировать мутации. Штамм дважды промывали 0,85% солевым раствором и после разбавления распределяли по минимальной среде, содержащей 1,7% агара, в которую добавляли вещество, подтверждающее резистентность клеток, в подходящей концентрации и в результате получали колонии. Каждую колонию культивировали в питательной среде и затем культивировали в посевной культуральной среде в течение 24 часов. После культивирования каждой колонии в ферментационной среде в течение 3-4 суток отбирали колонии, которые демонстрировали превосходное продуцирование ХМР, накапливаемого в культуральной среде. В частности, штаммы выбирали из тех, которые могут расти в среде, в которую добавлен фтортриптофан в соответствии с концентрациями (среда с добавкой), и более конкретно из тех, которые могут расти в среде с концентрацией фтортриптофана 100 мг/л и имеют повышенную концентрацию 5'- ксантиловой кислоты. Выбранный штамм был назван CJX1664.
- Минимальная среда: 20 г/л глюкозы, 1 г/л монокалия фосфата, 1 г/л дикалия фосфата, 2 г/л мочевины, 3 г/л сульфата аммония, 1 г/л сульфата магния, 100 мг/л хлорида кальция, 20 мг/л сульфата железа, 10 мг/л сульфата марганца, 10 мг/л сульфата цинка, 30 мкг/л биотина, 0,1 мг/л тиамина гидрохлорида, 0,8 мг/л сульфата меди, 20 мг/л аденина, 20 мг/л гуанина, рН 7,2.
- Среда с добавкой: среда, полученная в результате добавления фтортриптофана в концентрации 10 мг/л, 20 мг/л, 50 мг/л, 70 мг/л, 100 мг/л и 200 мг/л к минимальной среде.
Биохимические характеристики штамма CJX1664 приведены в таблице 12 ниже. Ссылаясь на таблицу 12, штамм CJX1664 может быть выращен в среде с добавкой, когда фтортриптофан добавлен в концентрации 100 мг/л.
Пример 5-2: Ферментативный титриметрический анализ CJX1664
После распределения посевной культуральной среды (2 мл) в аналитические пробирки (диаметр: 18 мм) пробирки автоклавировали. Каждый из АТСС6872 и CJX1664 инокулировали и инкубировали при 30°С в течение 24 ч при встряхивании и использовали в качестве посевной культуры. Ферментационную среду (29 мл) распределяли в каждую встряхиваемую колбу Эрленмейера объемом 250 мл и автоклавировали при 121°С в течение 15 минут. Посевную культуру (2 мл) инокулировали в среду и культивировали в течение 3 суток. Условия культивирования соответствовали 170 об/мин, 30°С и рН 7,5.
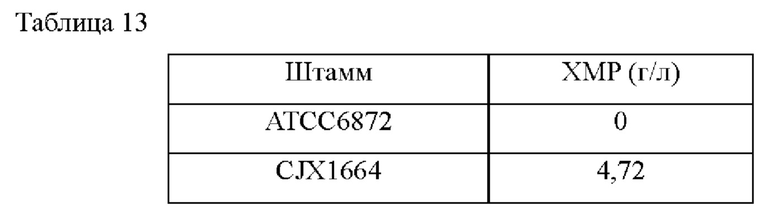
После завершения культивирования измеряли количество продуцируемого ХМР с помощью ВЭЖХ (SHIMADZU LC20A), и результаты культивирования были такими, как показано в Таблице 13 ниже.
Пример 5-3: Введение изменения в штамм CJX1664 и их оценка
Для подтверждения наличия изменения гена ригА штамма CJX1664, выбранного в Примере 5-1, хромосомную ДНК штамма CJX1664 амплифицировали с помощью ПЦР. В частности, сначала фрагменты purA амплифицировали с помощью ПЦР с использованием хромосомной ДНК штамма CJX1664 в качестве матрицы и праймеров SEQ ID NO: 17 и 18, при этом ПЦР проводили за 28 циклов денатурации при 94°С в течение 1 мин; отжига при 58°С в течение 30 с и полимеризации при 72°С в течение 2 мин с использованием ДНК-полимеразы Taq. Нуклеотидные последовательности амплифицированных фрагментов гена purA анализировали с использованием тех же праймеров, и в результате было подтверждено, что в гене purA штамма CJX1664 не было изменений.
Вектор, полученный в примере 4-3, трансформировали в штамм CJX1664, и штаммы, где вектор был встроен в геномную ДНК путем рекомбинации гомологичных последовательностей, отбирали на среде, содержащей 25 мг/л канамицина. Выбранные первичные штаммы подвергали вторичному кроссинговеру, и таким образом отбирали те штаммы, в которые был введен вариант целевого гена. Введение измененного гена в нужные трансформированные штаммы было подтверждено посредством анализа последовательности.
Оценивали продуцирование ХМР для каждого из штаммов CJX1664 и CJX1664::purA (G85S). После завершения культивирования количество ХМР, продуцированного каждым штаммом, измеряли методом с использованием ВЭЖХ, и результаты культивирования показаны в Таблице 14 ниже.
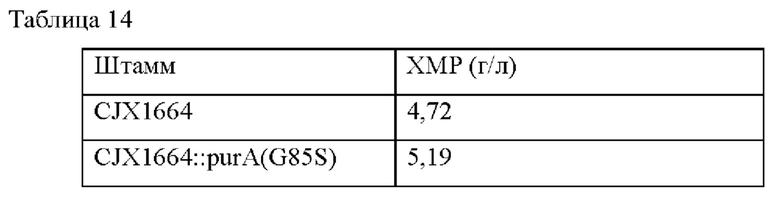
Как видно из приведенной выше Таблицы 14, штамм CJX1664::purA (G85S) продемонстрировал увеличение количества продуцируемого ХМР примерно на 109% по сравнению со штаммом CJX1664 (то есть ХМР-продуцирующим штаммом на основе АТСС6872).
Штамм CJX1664 был депонирован в Корейском центре культур микроорганизмов (КССМ) 6 июля 2018 года в соответствии с положениями Будапештского договора и ему присвоен регистрационный номер KCCM12285P. Кроме того, полученный штамм CJX1664::purA (G85S), также называемый CJX1665, был депонирован в KССМ 6 июля 2018 года в соответствии с положениями Будапештского договора и ему присвоен регистрационный номер KCCM12286P.
Пример 6: Замена аминокислоты другой аминокислотой в варианте purA
Пример 6-1: Получение вектора для введения аминокислоты в вариант purA
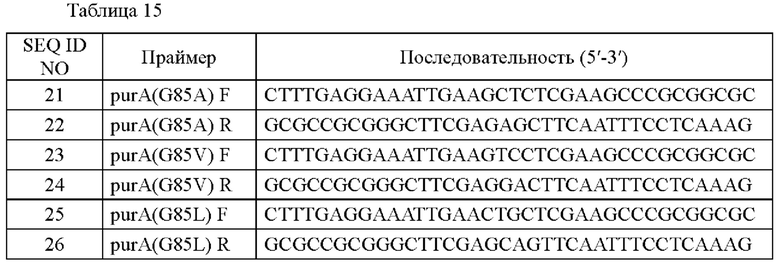
Посредством вышеуказанных примеров было подтверждено, что изменение purA(G85S) может улучшить продуцирование пуриновых нуклеотидов. В связи с этим, для подтверждения позиционной важности изменения purA исследовали влияние замены 85-й аминокислоты другой аминокислотой на продуцирование пуриновых нуклеотидов. Способ получения вектора для введения варианта purA (G85S) заключается в следующем. Сайт-направленный мутагенез выполняли с использованием вектора pDZ-purA (G85S), полученного в Примере 4, в качестве основы. В частности, ПЦР проводили с использованием последовательностей, показанных в таблице 15, в качестве праймеров в следующих условиях: 18 циклов денатурации при 94°С в течение 30 с, отжиг при 55°С в течение 30 с и удлинение при 68°С в течение 12 мин. Полученные ПЦР-продукты расщепляли DpnI, трансформировали в штамм DH5α и из него получали колонии. Плазмиды полученных таким образом колоний получали известным методом экстракции плазмид, и информация по полученным плазмидам представлена ниже в Таблице 15.

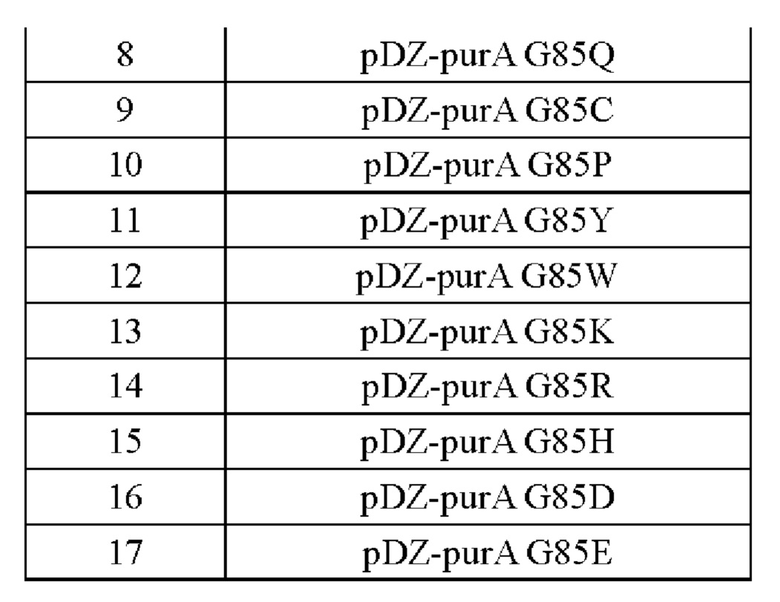
Пример 6-2: Получение штамма, в котором аминокислота заменена другой аминокислотой в соответствии с положением изменения варианта purA, н сравнение продуцирования 5'-инозиновой кислоты
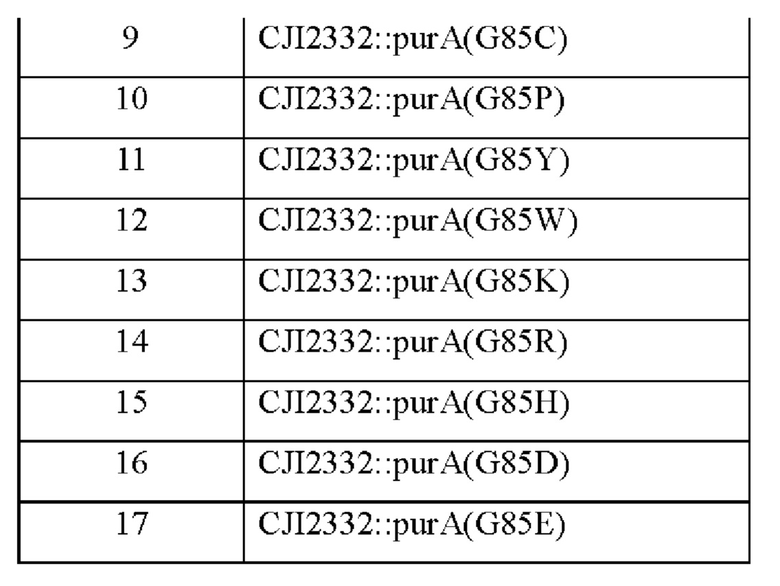
Каждый из 18 разновидностей векторов для введения вариантов, полученных в Примере 6-1, трансформировали в штамм CJI2332, и штаммы, где эти векторы были встроены в геномную ДНК посредством гомологичной рекомбинации, отбирали на среде, содержащей 25 мг/л канамицина. Отобранные первичные штаммы подвергали вторичному кроссинговеру, и таким образом отбирали те штаммы, в которые был введен вариант целевого гена. Для подтверждения введения варианта гена в нужные трансформированные штаммы проводили ПЦР с использованием праймеров SEQ ID NO: 17 и SEQ ID NO: 18, и продукты ПЦР подтверждали с помощью анализа последовательности. Штаммы были названы в соответствии с введенными вариантами, как показано в таблице 17.

Концентрацию 5'-инозиновой кислоты анализировали, культивируя штаммы таким же образом, как в примере 1.
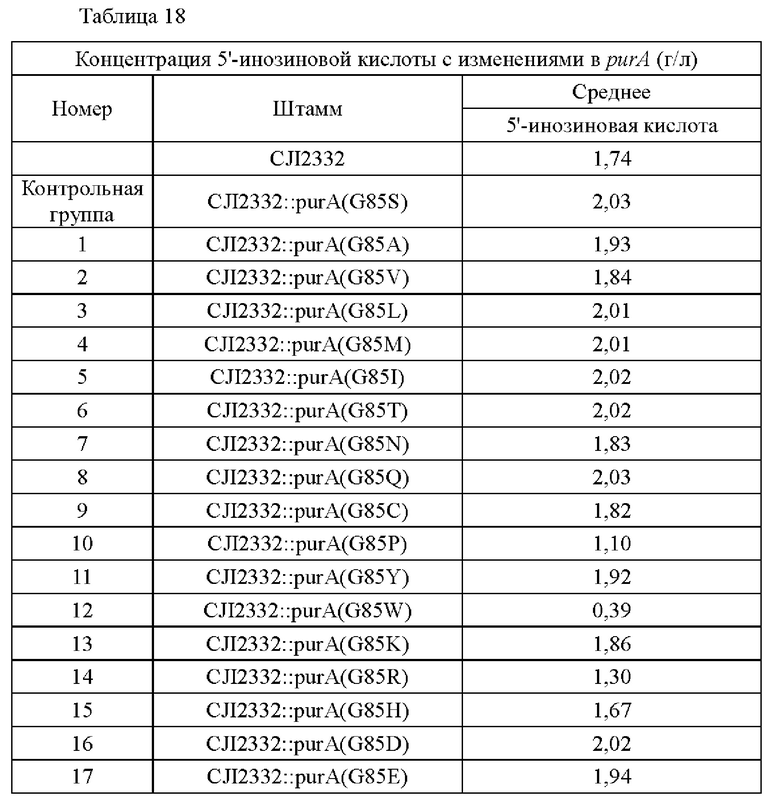
Обращаясь к приведенной выше таблице 18, было подтверждено, что штаммы, содержащие purA, в котором 85-я аминокислота аминокислотной последовательности, кодирующей ген purA, заменена другой аминокислотой, продемонстрировали значительное изменение количества продуцируемого IMP по сравнению с другими штаммами, которые не содержали вышеуказанное изменение. То есть было подтверждено, что 85-я аминокислота аминокислотной последовательности, кодирующей ген purA, является важным положением для изменения, связанного с продуцированием пуриновых нуклеотидов, и когда 85-я аминокислота аминокислотной последовательности, кодирующей ген purA, заменена аминокислотой, выбранной из группы, состоящей из серина, аланина, валина, лейцина, метионина, изолейцина, треонина, аспарагина, глутамина, цистеина, тирозина, лизина, аспарагиновой кислоты и глутаминовой кислоты, микроорганизм, имеющий такое изменение, может значительно повысить продуцирование пуриновых нуклеотидов.
Из вышеизложенного специалист в данной области техники, к которой относится настоящее изобретение, может понять, что настоящее изобретение может быть воплощено в других конкретных формах без изменения технической концепции или существенных характеристик настоящего изобретения. В этом отношении типичные воплощения, раскрытые в данном документе, представлены только для иллюстрации и не должны рассматриваться как ограничивающие объем настоящего изобретения. Напротив, предполагается, что настоящее изобретение охватывает не только типичные воплощения, но также различные альтернативы, модификации, эквиваленты и другие воплощения, которые могут быть включены в рамках сущности и объема настоящего раскрытия, как определено в прилагаемой формуле изобретения.
--->
<110> CJ CheilJedang Corporation
<120> Novel adenylosuccinate synthetase and method for producing purine
nucleotide using the same
<130> OPA18284
<150> 10-2018-0089855
<151> 2018-08-01
<160> 54
<170> KoPatentIn 3.0
<210> 1
<211> 1293
<212> DNA
<213> Unknown
<220>
<223> purA NT
<400> 1
atggcagcta tcgttatcgt cggcgcccaa tggggcgacg aaggcaaggg caaagctaca 60
gacatcctgg gtggacgcgt ggactacgtc gtcaagccca acggtggtaa caatgctggc 120
cacaccgttg tagtcggtgg cgaaaagtac gaactcaagc tgctgcccgc cggcatcctc 180
tccgaaaacg ccgtgccagt gctgggcaac ggcgtagtta tcaacctcga agcactcttt 240
gaggaaattg aaggcctcga agcccgcggc gctgatgctt cccgcttgcg tatttccgcc 300
aacgcgcacc tggttgcgcc ataccaccag accttagacc gcgtgcagga acgtttcttg 360
ggcaagcgcg cgattggcac caccggccgc ggtatcggcc cagcttatgc cgacaaggtc 420
gcgcgcgtgg gagttcgcgt gcaagacatc ttcgacgaat ccatcctgcg tcagaaggtc 480
gaatccgcgc tggatatcaa aaaccagatg ctggtcaaaa tgtacaaccg caaggcgatt 540
gaccctgaga ccatcgtcga atacttcctg tcctaccgcg accgcctcga gcctatggtc 600
gtggactccg agtacgagct caacaccgct ctggatgccg gcaagcacgt gctcatggaa 660
ggcggccagg ccaccatgct cgacgtggac cacggcacct acccattcgt gacctcgtcc 720
aacccaaccg ccggtggtgc atcggtaggc tctggcgtcg ggcctacccg catcacgcac 780
tccctgggca tcattaaggc gtacaccacc cgcgttggtg ctggcccatt cccaaccgag 840
ctctttgaca agtggggcga atacctccag accaccggcg gcgaggtcgg cgtcaacacc 900
ggccgcaccc gtcgttgtgg ctggtatgac tctgtcattg cacgctacgc atcgcgcgtc 960
aacggcttta ccgactactt ccttactaaa ctcgacgtgc tcaccggcat cggcgaaatc 1020
cctatttgcg tcgcctacga cgtcgatggc gaacgcttcg atgaaatgcc gctgactcag 1080
tcgcagtttc accacgcaca accaatttat gaaaccatgc cagcgtggga agaagacatc 1140
accggttgca ccacctttga ggagctgccg caaaaggcac aggattacgt gctgcggcta 1200
gaggagctgt ccggcacccg catgtcatac atcggcgttg gccctggccg tgaccagacc 1260
atcgtgcgcc acgacgtgct cgacgagaaa taa 1293
<210> 2
<211> 430
<212> PRT
<213> Unknown
<220>
<223> purA AA
<400> 2
Met Ala Ala Ile Val Ile Val Gly Ala Gln Trp Gly Asp Glu Gly Lys
1 5 10 15
Gly Lys Ala Thr Asp Ile Leu Gly Gly Arg Val Asp Tyr Val Val Lys
20 25 30
Pro Asn Gly Gly Asn Asn Ala Gly His Thr Val Val Val Gly Gly Glu
35 40 45
Lys Tyr Glu Leu Lys Leu Leu Pro Ala Gly Ile Leu Ser Glu Asn Ala
50 55 60
Val Pro Val Leu Gly Asn Gly Val Val Ile Asn Leu Glu Ala Leu Phe
65 70 75 80
Glu Glu Ile Glu Gly Leu Glu Ala Arg Gly Ala Asp Ala Ser Arg Leu
85 90 95
Arg Ile Ser Ala Asn Ala His Leu Val Ala Pro Tyr His Gln Thr Leu
100 105 110
Asp Arg Val Gln Glu Arg Phe Leu Gly Lys Arg Ala Ile Gly Thr Thr
115 120 125
Gly Arg Gly Ile Gly Pro Ala Tyr Ala Asp Lys Val Ala Arg Val Gly
130 135 140
Val Arg Val Gln Asp Ile Phe Asp Glu Ser Ile Leu Arg Gln Lys Val
145 150 155 160
Glu Ser Ala Leu Asp Ile Lys Asn Gln Met Leu Val Lys Met Tyr Asn
165 170 175
Arg Lys Ala Ile Asp Pro Glu Thr Ile Val Glu Tyr Phe Leu Ser Tyr
180 185 190
Arg Asp Arg Leu Glu Pro Met Val Val Asp Ser Glu Tyr Glu Leu Asn
195 200 205
Thr Ala Leu Asp Ala Gly Lys His Val Leu Met Glu Gly Gly Gln Ala
210 215 220
Thr Met Leu Asp Val Asp His Gly Thr Tyr Pro Phe Val Thr Ser Ser
225 230 235 240
Asn Pro Thr Ala Gly Gly Ala Ser Val Gly Ser Gly Val Gly Pro Thr
245 250 255
Arg Ile Thr His Ser Leu Gly Ile Ile Lys Ala Tyr Thr Thr Arg Val
260 265 270
Gly Ala Gly Pro Phe Pro Thr Glu Leu Phe Asp Lys Trp Gly Glu Tyr
275 280 285
Leu Gln Thr Thr Gly Gly Glu Val Gly Val Asn Thr Gly Arg Thr Arg
290 295 300
Arg Cys Gly Trp Tyr Asp Ser Val Ile Ala Arg Tyr Ala Ser Arg Val
305 310 315 320
Asn Gly Phe Thr Asp Tyr Phe Leu Thr Lys Leu Asp Val Leu Thr Gly
325 330 335
Ile Gly Glu Ile Pro Ile Cys Val Ala Tyr Asp Val Asp Gly Glu Arg
340 345 350
Phe Asp Glu Met Pro Leu Thr Gln Ser Gln Phe His His Ala Gln Pro
355 360 365
Ile Tyr Glu Thr Met Pro Ala Trp Glu Glu Asp Ile Thr Gly Cys Thr
370 375 380
Thr Phe Glu Glu Leu Pro Gln Lys Ala Gln Asp Tyr Val Leu Arg Leu
385 390 395 400
Glu Glu Leu Ser Gly Thr Arg Met Ser Tyr Ile Gly Val Gly Pro Gly
405 410 415
Arg Asp Gln Thr Ile Val Arg His Asp Val Leu Asp Glu Lys
420 425 430
<210> 3
<211> 1500
<212> DNA
<213> Unknown
<220>
<223> purF
<400> 3
gtggtgaaca ctactttccc cagcgacgtg aatttagatg accaaggcga gcaagaaccc 60
cgcgaagagt gcggtgtctt tggcgtctgg gctcctggtg aagatgttgc gacactgacc 120
tactttggtc tgttcgcatt gcagcatcgt gggcaggaag ctgcaggtat cggcgtcggt 180
gatggagacc gcctcgttgt cttcaaagac atgggcttgg tctcgaatat tttcgatgag 240
tccattttaa attccctcca tggctccgtg ggcgtggggc atacgcgcta ctcgactgcc 300
ggtggcaaag agtggtcgaa tgtccagccg atgtttaata ccacctcaaa tggggtagac 360
atcgctttgt gccacaacgg caacttggtg aactaccaag aactgcgcga tgaagcagta 420
gctctgggac tttaccgaga gaatgaaaaa tccctgtcgg attccatgat catgacagct 480
ttgctggcgc acggagtcgg ggaaggcaac tctgtctttg acgccgctaa gcaactgctg 540
ccaagcatca aaggcgcttt ttgcttgacc tttaccgatg gcaagacctt gtacgccgcg 600
cgtgacccgc acggtgtacg ccccttggtc attggccgct tggcgcaagg ctgggttgtt 660
gcttccgaaa cctgtgcgct ggatatcgtg ggcgcacagt ttatccgtga ggtagagccc 720
ggtgaactta tctctgtcaa tgaggcagga atccacagcg aaaaattcgc tgagccgaag 780
cgccagggct gcgtctttga atacgtctac ttggcacgtc cagacaccgt gatcaaaggc 840
cgcaacgttc acgcgacgcg cgtggatatt ggtcgcgcac ttgcgaaatc tcaccctgcg 900
ccagaagctg acatggtcat ccccgtgcca gaatccggaa acccggcagc tgttggctac 960
gcccgggaat cgggcctgac atttgcgcac ggcttggtca aaaacgccta cgtgggtcga 1020
accttcattc agcccaccca gaccttgcgc cagctgggta ttcgcctcaa gctcaacccc 1080
ctgcgcgagg tcatcgaggg caagtcactc gttgttgtag atgactctat tgtccgcggc 1140
aacacccaac gcgcgctggt gcgcatgctg cgtgaagcag gcgctgctga agtgcacgtg 1200
cgcattgctt caccgccagt caaatggcct tgtttctacg gcattgactt cgcctcgcct 1260
ggtgaattga ttgctaatat caagccttct gatgatcctc aggtagtaac cgatgcagtg 1320
tgcgaagcta tcggagcaga ctctttaggg tttgtatctg tagatgagat ggttgaggca 1380
acgcaccaac ctatcaattc cttgtgtacc gcttgctttg atggcaacta cgaactcgga 1440
cttccgaccg ctaaccccaa tgctgacgct gtgcgaactt tgctcagcca aaagaactga 1500
1500
<210> 4
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> purF g1a-1
<400> 4
gctctagacc actctaagac gcggccacc 29
<210> 5
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> purF g1a-2
<400> 5
aagtagtgtt caccatgacg ctgattctac taagttt 37
<210> 6
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> purF g1a-3
<400> 6
agtagaatca gcgtcatggt gaacactact ttccccag 38
<210> 7
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> purF g1a-4
<400> 7
gctctagact gtgcgcccac gatatccag 29
<210> 8
<211> 1521
<212> DNA
<213> Unknown
<220>
<223> guaB
<400> 8
atgaccgaaa atcgtgtttc taccggtgga gatgacccaa ataaggttgc attgcatggc 60
ttgacgtttg atgacgtgct gctgctacct gccgaatcca atgttgttcc gtcggaagta 120
gacacttcgg cgcagttcac ccgcaatact cgtttaggta ttcctttggc atcggctgcg 180
atggacacgg ttactgaggc gcgcatggct attgccatgg cacgccaggg tggcattggt 240
gtcttgcacc gcaacttgtc ctcgcaagag caggcggagc aggtcgaaat cgtcaagcgc 300
tctgagtccg gcatggtcac cgaccctgtg accgcgaatc cagacatgac tatccaggaa 360
gttgatgacc tgtgtgcacg cttccgcatc tctggtcttc ctgtggtcaa cgaagacggc 420
accttgttgg gcatttgcac caaccgcgat atgcgctttg agcgcgacta ttcccgcaag 480
gtttctgaca tcatgaccgc tatgccgctg gttgtggcaa aagaaggcgt cagcaaggaa 540
gaagccctgg atctgctgtc gacgaacaag gtagaaaagc tacctatcgt tgataaaaac 600
aacaagctgg tcggtctgat taccgttaaa gactttgtta agaccgaaca gttcccgaat 660
tcctccaagg atgcttcggg ccgcttgcta gtagcagcag gtattggtac cggcgaggag 720
tcttatgagc gtgcaggctt gcttgtcgat gccggcgtgg acgttctcat tgtcgactcc 780
gcacacgcgc acaataaccg cgtgctggaa atggtctcgc gcgtcaagaa tgacttcggc 840
tccaagattg atgttgtcgg cggcaacctg gcaacacgct cggcagcaaa ggcgatgatt 900
gaggctggcg cagacgccat caaggtgggt attggtcctg gttctatctg caccacccgt 960
gtggttgctg gtgttggtgc accacagatt accgcgatca tggaagcagc taccgtggct 1020
tctgctgcgg gcgtgccttt gattgcagac ggcggcatgc agtactccgg tgacgttgct 1080
aaggctttgg ctgctggcgc ggactcggtc atgctgggct cgatgttcgc aggcaccctg 1140
gaggctcctg gtgacatcgt gattgtcggc ggcaagcagt acaagcgcta ccgcggcatg 1200
ggttcgatgg gcgctatgca aggccgtggc ctctccggcg agaagcgttc ttactccaag 1260
gaccgctact tccaggcaga tgtgcgcagc gaagataagc tggttccaga aggcgtggaa 1320
ggcaaggttc cttaccgcgg cgaaattggt cagattaccc accagattgt gggcggtttg 1380
cgcgcggcaa tgggctacac tggctccgct actattgaag agctgaagac caagcagttc 1440
gtgcgtatta ccactgctgg cttggctgag tcgcacccgc accacctgca gcaaactgta 1500
gaagctccga actaccgtta a 1521
<210> 9
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> guaB a1t-1
<400> 9
gctctagact acgacaacac ggtgcctaa 29
<210> 10
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> guaB a1t-2
<400> 10
cacgattttc ggtcaatacg ggtcttctcc ttcgcac 37
<210> 11
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> guaB a1t-3
<400> 11
aggagaagac ccgtattgac cgaaaatcgt gtttct 36
<210> 12
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> guaB a1t-4
<400> 12
gctctagaat cgacaagcaa gcctgcacg 29
<210> 13
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> purA 5' праймер F (матрич.)
<400> 13
atggctaaat acattatcac t 21
<210> 14
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> purA 5' праймер R (матрич.)
<400> 14
tgtgctggag acccctcata g 21
<210> 15
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> purA праймер F допускающей ошибки ПЦР
<400> 15
aagggcaaag ctacagacat c 21
<210> 16
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> purA праймер R допускающей ошибки ПЦР
<400> 16
ccgccgagca tgagaacccg a 21
<210> 17
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> purA seq F
<400> 17
gacgcgtcgg aatcgccgat a 21
<210> 18
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> purA seq R
<400> 18
ccgccgagca tgagaacccg a 21
<210> 19
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85S) F
<400> 19
gctctagatg ccggcattt ttcgaagc 27
<210> 20
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85S) R
<400> 20
gctctagaaa gtagtcggta aagccgttg 29
<210> 21
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85A) F
<400> 21
ctttgaggaa attgaagctc tcgaagcccg cggcgc 36
<210> 22
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85A) R
<400> 22
gcgccgcggg cttcgagagc ttcaatttcc tcaaag 36
<210> 23
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85V) F
<400> 23
ctttgaggaa attgaagtcc tcgaagcccg cggcgc 36
<210> 24
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85V) R
<400> 24
gcgccgcggg cttcgaggac ttcaatttcc tcaaag 36
<210> 25
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85L) F
<400> 25
ctttgaggaa attgaactgc tcgaagcccg cggcgc 36
<210> 26
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85L) R
<400> 26
gcgccgcggg cttcgagcag ttcaatttcc tcaaag 36
<210> 27
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85M) F
<400> 27
ctttgaggaa attgaaatgc tcgaagcccg cggcgc 36
<210> 28
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85M) R
<400> 28
gcgccgcggg cttcgagcat ttcaatttcc tcaaag 36
<210> 29
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85I) F
<400> 29
ctttgaggaa attgaaatcc tcgaagcccg cggcgc 36
<210> 30
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85I) R
<400> 30
gcgccgcggg cttcgaggat ttcaatttcc tcaaag 36
<210> 31
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85T) F
<400> 31
ctttgaggaa attgaaactc tcgaagcccg cggcgc 36
<210> 32
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85T) R
<400> 32
gcgccgcggg cttcgagagt ttcaatttcc tcaaag 36
<210> 33
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85N) F
<400> 33
ctttgaggaa attgaaaacc tcgaagcccg cggcgc 36
<210> 34
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85N) R
<400> 34
gcgccgcggg cttcgaggtt ttcaatttcc tcaaag 36
<210> 35
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85Q) F
<400> 35
ctttgaggaa attgaacagc tcgaagcccg cggcgc 36
<210> 36
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85Q) R
<400> 36
gcgccgcggg cttcgagctg ttcaatttcc tcaaag 36
<210> 37
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85C) F
<400> 37
ctttgaggaa attgaatgcc tcgaagcccg cggcgc 36
<210> 38
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85C) R
<400> 38
gcgccgcggg cttcgaggca ttcaatttcc tcaaag 36
<210> 39
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85P) F
<400> 39
ctttgaggaa attgaaccac tcgaagcccg cggcgc 36
<210> 40
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85P) R
<400> 40
gcgccgcggg cttcgagtgg ttcaatttcc tcaaag 36
<210> 41
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85Y) F
<400> 41
ctttgaggaa attgaatacc tcgaagcccg cggcgc 36
<210> 42
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85Y) R
<400> 42
gcgccgcggg cttcgaggta ttcaatttcc tcaaag 36
<210> 43
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85W) F
<400> 43
ctttgaggaa attgaatggc tcgaagcccg cggcgc 36
<210> 44
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85W) R
<400> 44
gcgccgcggg cttcgagcca ttcaatttcc tcaaag 36
<210> 45
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85K) F
<400> 45
ctttgaggaa attgaaaagc tcgaagcccg cggcgc 36
<210> 46
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85K) R
<400> 46
gcgccgcggg cttcgagctt ttcaatttcc tcaaag 36
<210> 47
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85R) F
<400> 47
ctttgaggaa attgaacgcc tcgaagcccg cggcgc 36
<210> 48
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85R) R
<400> 48
gcgccgcggg cttcgaggcg ttcaatttcc tcaaag 36
<210> 49
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85H) F
<400> 49
ctttgaggaa attgaacacc tcgaagcccg cggcgc 36
<210> 50
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85H) R
<400> 50
gcgccgcggg cttcgaggtg ttcaatttcc tcaaag 36
<210> 51
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85D) F
<400> 51
ctttgaggaa attgaagatc tcgaagcccg cggcgc 36
<210> 52
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85D) R
<400> 52
gcgccgcggg cttcgagatc ttcaatttcc tcaaag 36
<210> 53
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85E) F
<400> 53
ctttgaggaa attgaagaac tcgaagcccg cggcgc 36
<210> 54
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> purA(G85E) R
<400> 54
gcgccgcggg cttcgagttc ttcaatttcc tcaaag 36
<---
Настоящее изобретение относится к биотехнологии, в частности к варианту аденилосукцинат-синтетазы, к микроорганизму, содержащему его, и к способу получения пуриновых нуклеотидов с использованием такого микроорганизма. Изобретение позволяет с высокой степенью эффективности получать пуриновые нуклеотиды. 7 н. и 5 з.п. ф-лы, 18 табл., 6 пр.
1. Вариант аденилосукцинат-синтетазы, обладающий активностью аденилосукцинат-синтетазы, где 85-я аминокислота с N-конца аминокислотной последовательности SEQ ID NO: 2 заменена другой аминокислотой.
2. Вариант аденилосукцинат-синтетазы по п.1, где другая аминокислота выбрана из группы, состоящей из серина, аланина, валина, лейцина, метионина, изолейцина, треонина, аспарагина, глутамина, цистеина, тирозина, лизина, аспарагиновой кислоты и глутаминовой кислоты.
3. Полинуклеотид, кодирующий вариант аденилосукцинат-синтетазы по п. 1.
4. Экспрессионный вектор, содержащий полинуклеотид по п.3.
5. Микроорганизм рода Corynebacterium, продуцирующий пуриновые нуклеотиды посредством содержащегося в нем варианта аденилосукцинат-синтетазы по п. 1 или вектора по п. 4.
6. Микроорганизм по п. 5, где микроорганизм рода Corynebacterium представляет собой Corynebacterium stationis.
7. Способ получения 5’-инозинмонофосфата, включающий: культивирование микроорганизма рода Corynebacterium по п. 5 в среде; и выделение 5’-инозинмонофосфата из микроорганизма или из среды.
8. Способ по п. 7, где микроорганизм рода Corynebacterium представляет собой Corynebacterium stationis.
9. Способ получения 5’-ксантозинмонофосфата, включающий:
культивирование микроорганизма рода Corynebacterium по п. 5 в среде и выделение 5’-ксантозинмонофосфата из микроорганизма или из среды.
10. Способ по п. 9, где микроорганизм рода Corynebacterium представляет собой
Corynebacterium stationis.
11. Способ получения 5’-гуанинмонофосфата, включающий:
культивирование микроорганизма рода Corynebacterium по п. 5 в среде и выделение 5’-гуанинмонофосфата из микроорганизма или из среды.
12. Способ по п. 11, где микроорганизм рода Corynebacterium представляет собой Corynebacterium stationis.
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Механическая топочная решетка с наклонными частью подвижными, частью неподвижными колосниковыми элементами | 1917 |
|
SU1988A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Пуговица для прикрепления ее к материи без пришивки | 1921 |
|
SU1992A1 |
Авторы
Даты
2022-04-18—Публикация
2018-08-23—Подача