Область изобретения
Настоящая заявка на изобретение относится к варианту альдолазы aroG (фосфо-2-дегидро-3-дезоксигептаноат альдолазы) и способу получения аминокислот с его использованием.
Предшествующий уровень техники
L-аминокислоты являются основными строительными блоками белков и используются в качестве важных материалов для фармацевтического сырья, пищевых добавок, кормов для животных, пищевых добавок, пестицидов, бактерицидов и т.д. Поэтому промышленное производство аминокислот стало важным промышленным процессом с экономической точки зрения.
Были проведены различные исследования для эффективной продукции аминокислот; например, были предприняты усилия разработать микроорганизмы или технологии процесса ферментации для продукции аминокислот с высокой эффективностью. В частности, были разработаны специфические подходы к материалам-мишеням, такие как усиление экспрессии генов, кодирующих ферменты, вовлеченные в биосинтез аминокислот в штаммах рода Corynebacterium, или делеция генов, не являющихся необходимыми для биосинтеза аминокислот (US 9109242 В2). Дополнительно к этим способам также использовали способ удаления генов, которые не вовлечены в продукцию аминокислот, и способ удаления генов, функции которых для продукции аминокислот не были конкретно известны.
Между тем, среди аминокислот аминокислоты с разветвленной цепью относятся к трем аминокислотам валину, лейцину и изолейцину, и известно, что они главным образом подвергаются метаболизму в мышечной ткани и служат источником энергии во время физических нагрузок. Поскольку известно, что аминокислоты с разветвленной цепью играют важную роль в поддержании мышц и их роста во время физических нагрузок, их использование увеличивается. Тем не менее, поскольку в пути биосинтеза аминокислот с разветвленной цепью образуется большое количество побочных продуктов, важно уменьшить количество побочных продуктов для получения аминокислот с разветвленной цепью с высоким выходом и с высокой чистотой.
Описание изобретения
Техническая проблема
Авторы настоящего изобретения подтвердили то, что микроорганизм, в который введен вновь разработанный вариант альдолазы aroG, может продуцировать аминокислоты с разветвленной цепью с высоким выходом и высокой чистотой, таким образом, завершая настоящую заявку на изобретение.
Техническое решение
Одна из задач настоящей заявки на изобретение заключается в том, чтобы предложить вариант альдолазы aroG (фосфо-2-дигидро-3-дезоксигептаноат альдолаза), в котором любая одна или более чем одна аминокислота, соответствующая положениям 217, 310, 403 и 462 от N-конца в аминокислотной последовательности SEQ ID NO: 1, заменена на другую аминокислоту.
Еще одна задача настоящей заявки на изобретение заключается в том, чтобы предложить полинуклеотид, кодирующий вариант, и содержащий его вектор.
Еще одна задача настоящей заявки на изобретение заключается в том, чтобы предложить микроорганизм рода Corynebacterium, включающий одно или более чем одно из следующего: вариант альдолазы aroG, полинуклеотид и вектор.
Еще одна задача настоящей заявки на изобретение заключается в том, чтобы предложить способ получения аминокислот с разветвленной цепью, включающий культивирование указанного микроорганизма в среде.
Благоприятные эффекты
Когда используют вариант альдолазы aroG в соответствии с настоящей заявкой на изобретение, тогда продукция побочных продуктов может быть уменьшена, и аминокислоты с разветвленной цепью можно получать с высоким выходом по сравнению с тем случаем, когда не используют вариант альдолазы aroG.
Подробное описание предпочтительных воплощений
Описание настоящего изобретения подробно будет описано далее. Между тем, каждое описание и воплощение, раскрытые в этом описании изобретения, также можно применять соответственно к другим описаниям и воплощениям. То есть все комбинации различных элементов, раскрытых в этом описании изобретения, входят в объем настоящей заявки на изобретение. Кроме того, объем настоящей заявки на изобретение не ограничен конкретным описанием, представленным ниже.
Кроме того, специалистам в данной области техники понятно или они могут подтвердить с использованием не более чем стандартных экспериментов множество эквивалентов конкретных аспектов описанного здесь изобретения. Кроме того, предполагается, что эти эквиваленты должны быть включены в настоящую заявку на изобретение.
В одном из аспектов настоящей заявки на изобретение предложен вариант альдолазы aroG (фосфо-2-дегидро-3-дезоксигептаноат альдолазы), в котором любая одна или более чем одна из аминокислот, соответствующая положениям 217, 310, 403 и 462 от N-конца в аминокислотной последовательности SEQ ID NO: 1 заменена на другую аминокислоту.
Вариант альдолазы aroG относится к варианту, в котором любая одна или более чем одна из аминокислот, соответствующих положениям 217, 310, 403 и 462 от N-конца в аминокислотной последовательности SEQ ID NO: 1 заменена на другую аминокислоту, в полипептиде, обладающим активностью альдолазы aroG, или в альдолазе aroG
Используемый здесь термин «альдолаза aroG» представляет собой фермент, который обладает способностью катализировать следующую реакцию:
Фосфоэнолпируват + D-эритрозо-4-фосфат + H2O ↔ 3-дезокси-D-арабино-гепт-2-улозонат-7-фосфат + фосфат
Альдолаза aroG в соответствии с настоящей заявкой на изобретение может представлять собой альдолазу aroG, в отношении которой применены модификации для получения предложенного здесь варианта альдолазы aroG, или полипептид, обладающий активностью альдолазы aroG. В частности, он может представлять собой встречающийся в природе полипептид или полипептид дикого типа, может представлять собой его зрелый полипептид, и может без ограничения включать вариант или его функциональный фрагмент до тех пор, пока он является родительским для варианта альдолазы aroG в соответствии с настоящей заявкой на изобретение.
Использованная здесь альдолаза aroG может представлять собой полипептид с SEQ ID NO: 1, но не ограничиваться этим. В одном из воплощений это может быть полипептид, обладающий идентичностью последовательности приблизительно 60%, 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% или более чем 99% с полипептидом с SEQ ID NO: 1, и любой полипептид, обладающий активностью, идентичной или соответствующей полипептиду, состоящему из аминокислотной последовательности SEQ ID NO: 1, без ограничения может быть включен в объем альдолазы aroG.
Последовательность альдолазы aroG в соответствии с настоящей заявкой на изобретение может быть получена из GenBank в NCBI (Национальный центр биотехнологической информации), представляющего собой известную базу данных. В частности, он может представлять собой полипептид, кодируемый геном aroG, но не ограничиваться этим.
Используемый здесь термин «вариант» относится к полипептиду, имеющему одну или более чем одну аминокислоту, отличающуюся от аминокислотной последовательности варианта перед модификацией путем консервативной замены и/или таких модификаций, что функции или свойства полипептида сохраняются. Такие варианты как правило могут быть идентифицированы путем модификации одной или более чем одной из аминокислотных последовательностей полипептида и путем оценки свойств модифицированного полипептида. Иначе говоря, способность вариантов может быть увеличена, не изменена или уменьшена по сравнению с полипептидом до модификации. Кроме того, некоторые варианты могут включать варианты, в которых одна или более чем одна часть, такая как N-концевая лидерная последовательность или трансмембранный домен, удалены. Другие варианты могут включать варианты, в которых фрагмент удален из N- и/или С-конца зрелого белка. Термин «вариант» может быть использован взаимозаменяемо с терминами, такими как модифицированный, модификация, модифицированный полипептид, модифицированный белок, мутация и мутант и т.п., без ограничения ими при условии, что термины используют для обозначения вариации.
Кроме того, варианты также могут включать делецию или вставку аминокислоты, которые обладают минимальным влиянием на свойства и вторичную структуру полипептида. Например, по N-концу варианта котрансляционно или посттрансляционно может быть конъюгирована сигнальная (или лидерная) последовательность, которая вовлечена в перенос белков. Кроме того, варианты могут быть также конъюгированы с другой последовательностью или линкером для идентификации, очистки или синтеза полипептида.
Предложенный здесь вариант может представлять собой вариант альдолазы aroG, в котором любая одна или более чем одна аминокислота, соответствующая положениям 217, 310, 403 и 462 от N-конца в аминокислотной последовательности SEQ ID NO: 1, заменена на другую аминокислоту. Например, в вышеприведенных положениях 2 или более, 3 или более, или все 4 аминокислоты могут быть заменены, но не ограничиваться этим.
Аминокислота, соответствующая положению 217 от N-конца в аминокислотной последовательности SEQ ID NO: 1, может представлять собой аргинин; аминокислота, соответствующая положению 310, может представлять собой лизин; аминокислота, соответствующая положению 403, может представлять собой аргинин; и/или аминокислота, соответствующая положению 462, может представлять собой глутаминовую кислоту.
Предложенный здесь вариант может включать одну или более чем одну замену из: замены аминокислоты, соответствующей положению 217 от N-конца в аминокислотной последовательности SEQ ID NO: 1, на аминокислоту за исключением аргинина; замены аминокислоты, соответствующей положению 310, на аминокислоту за исключением лизина; замены аминокислоты, соответствующей положению 403, на аминокислоту за исключением аргинина; и замены аминокислоты, соответствующей положению 462, на аминокислоту за исключением глутаминовой кислоты, но не ограничиваться этим.
Термин «другая аминокислота» не ограничен до тех пор, пока он представляет собой аминокислоту, отличающуюся от аминокислоты перед заменой. Тем не менее, когда указывается то, что «конкретная аминокислота была заменена», тогда очевидно то, что аминокислота заменена на аминокислоту, отличающуюся от аминокислоты перед заменой, даже если специально не указано то, что аминокислота заменена на отличающуюся аминокислоту.
В одном из воплощений вариант в соответствии с настоящей заявкой на изобретение может быть таким, в котором любая одна или более чем одна аминокислота, соответствующая положениям 217, 310, 403 и 462 в аминокислотной последовательности SEQ ID NO: 1, которая представляет собой референсный белок, заменена на гидрофобную аминокислоту или алифатическую аминокислоту, которая отличается от аминокислоты перед заменой.
В частности, вариант может быть таким, в котором любая одна или более чем одна аминокислота, соответствующая положениям 217, 310, 403 и 462 в аминокислотной последовательности SEQ ID NO: 1, заменена на гидрофобную (неполярную) аминокислоту или алифатическую аминокислоту. Алифатическая аминокислота может представлять собой, например, аминокислоту, выбранную из группы, состоящей из глицина, аланина, валина, лейцина и изолейцина, но не ограничивается ими. Гидрофобная (неполярная) аминокислота может представлять собой, например, аминокислоту, выбранную из группы, состоящей из глицина, метионина, аланина, валина, лейцина, изолейцина, пролина, фенилаланина, тирозина и триптофана, но не ограничивается ими.
В одном из воплощений вариант в соответствии с настоящей заявкой на изобретение может представлять собой вариант, в котором любая одна или более чем одна аминокислота, соответствующая положениям 217, 310, 403 и 462 в аминокислотной последовательности SEQ ID NO: 1, заменена на аминокислоту, отличающуюся от аминокислоты перед заменой среди аминокислот малого размера, но не ограничивается ими.
Используемый здесь термин «аминокислота малого размера» может включать аминокислоту с относительно небольшим размером среди 20 аминокислот, такую как глицин, аланин, серин, треонин, цистеин, валин, лейцин, изолейцин, пролин и аспарагин, и конкретно может относиться к глицину, аланину, серину, треонину, цистеину, валину, лейцину, изолейцину и пролину но не ограничивается ими. Конкретней, он может относиться к глицину, аланину, валину, лейцину, изолейцину, серину и треонину, но не ограничивается ими.
Конкретнее, замена на еще одну аминокислоту в отношении варианта в настоящей заявке на изобретение может представлять собой замену на аланин, но не ограничивается ими.
Используемый здесь термин «соответствующий» относится к аминокислотному остатку в положении, указанному в полипептиде, или аминокислотному остатку, который похож, идентичен или гомологичен остатку, указанному в полипептиде. Идентификация аминокислоты в соответствующем положении может определять конкретную аминокислоту в последовательности, которая относится к специфической последовательности. Используемый здесь термин «соответствующая область» обычно относится к похожему или соответствующему положению в родственном белке или референсном белке.
Например, любая аминокислотная последовательность выравнивается с SEQ ID NO: 1, и на основании этого выравнивания каждый аминокислотный остаток аминокислотной последовательности может быть пронумерован со ссылкой на номер положения аминокислотного остатка, соответствующего аминокислотному остатку в SEQ ID NO: 1. Например, алгоритм выравнивания последовательности, такой как описанный здесь, может определять положение аминокислоты или положение, в котором осуществляют модификации, такие как замены, вставки или делеции, путем сравнения с интересующей последовательностью (также названной как «референсная последовательность»).
Пример выравнивания может быть определен при помощи алгоритма Нидлмана-Вунша (Needleman and Wunsch, 1970, J. Mol. Biol. 48:443-453), который осуществляют с использованием программы Needleman в пакете EMBOSS (EMBOSS: The European Molecular Biology Open Software Suite, Rice et al., 2000, Trends Genet. 16:276-277) и т.п., но не ограничиваться ими, и подходящим образом могут быть использованы программы для выравнивания последовательностей, такие как алгоритмы попарного сравнения последовательностей, и т.п., известные в области техники.
В одном из воплощений вариант в соответствии с настоящей заявкой на изобретение может быть таким, в котором любая одна или более чем одна аминокислота, соответствующая положениям 217, 310, 403 и 462 в аминокислотной последовательности SEQ ID NO: 1, заменена на другую аминокислоту, в то же время, обладая идентичностью последовательности приблизительно 60%, 70%, 75%. 80%. 85%, 90%, 95%, 96%, 97%, 98% или 99% или более чем 99% с полипептидом с SEQ ID NO: 1.
В одном из воплощений вариант в соответствии с настоящей заявкой на изобретение может включать аминокислотную последовательность, обладающую гомологией или идентичностью по меньшей мере 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 99,5%, 99,7% или 99,9% с аминокислотной последовательностью, представленной в SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 23 или SEQ ID NO: 25.
В частности, вариант в соответствии с настоящей заявкой на изобретение может иметь, включать, состоять из или по существу состоять из аминокислотной последовательности, представленной в SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 23 или SEQ ID NO: 25.
В одном из воплощений вариант в соответствии с настоящей заявкой на изобретение может включать аминокислотную последовательность, основанную на аминокислотной последовательности SEQ ID NO: 1, в которой любая одна или более чем одна аминокислота, соответствующая положениям 217, 310, 403 и 462, представляет собой аланин, и которая имеет гомологию или идентичность по меньшей мере 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 99.5%, 99,7% или 99,9% с аминокислотной последовательностью, представленной в SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 23 или SEQ ID NO: 25. Кроме того, понятно то, что варианты, имеющие аминокислотную последовательность, в которой часть аминокислотной последовательности удалена, модифицирована, заменена, консервативно заменена или добавлена, могут попадать в объем настоящей заявки на изобретение до тех пор, пока аминокислотная последовательность обладает такой гомологией или идентичностью и демонстрирует эффективность, соответствующую варианту, раскрытому в настоящей заявке на изобретение.
Например, он может включать добавки или делеции последовательности, природные мутации, молчащие мутации или консервативные замены, которые не изменяют функцию варианта настоящей заявки на изобретение по N-концу, по С-концу, и/или в аминокислотной последовательности.
Используемый здесь термин «консервативная замена» относится к замене аминокислоты на другую аминокислоту, обладающую похожими структурными и/или химическими свойствами. Такая аминокислотная замена как правило может осуществляться на основе сходства полярности, заряда, растворимости, гидрофобности, гидрофильности и/или амфипатической природы остатка. Как правило, консервативные замены могут обладать не большим действием или отсутствием действия в отношении активности белка или полипептида.
Использованный здесь термин «гомология» или «идентичность» относится к степени сходства между двумя данными аминокислотными последовательностями или нуклеотидными последовательностями, и она может быть выражена в процентах. Часто термины «гомология» и «идентичность» можно использовать взаимозаменяемо друг с другом.
Гомологию или идентичность последовательностей консервативных полипептидных или полинуклеотидных последовательностей можно определить путем стандартных алгоритмов выравнивания, и можно использовать вместе с штрафом за пропуск в последовательности, установленный по умолчанию в используемой программе. По существу, как правило ожидают, что гомологичные или идентичные последовательности могут гибридизоваться с целой последовательностью или частью последовательности в умеренно строгих или очень строгих условиях. Понятно то, что при гибридизации полинуклеотидов также рассматривают полинуклеотиды, которые содержат вырожденные кодоны вместо кодонов.
Имеют ли любые две полинуклеотидные последовательности гомологию, сходство или идентичность может быть, например, определено с использованием известного компьютерного алгоритма, такого как программа «FASTA», с использованием параметров по умолчанию (Pearson et at, (1988) Proc. Natl. Acad. Sci. USA 85:2444). Альтернативно, гомология, сходство или идентичность могут быть определены с использованием алгоритма Нидлмана-Вунша (Needleman and Wunsch, 1970, J. Mot Biol. 48:443-453), который осуществляют с использованием программы Needleman пакета EMBOSS (EMBOSS:The European Molecular Biology Open Software Suite, Rice et at, 2000, Trends Genet. 16:276-277) (версия 5.0.0 или более поздняя) (программный пакет GCG (Devereux, J., et at, Nucleic Acids Research 12:387 (1984)), BLASTP, BLASTN, FASТА (Atschul, S.F., et at, J MOLEC BIOL 215:403 (1990); Guide to Huge Computers, Martin J. Bishop, ed., Academic Press, San Diego, 1994, и CARILLO et at (1988) SIAM J Applied Math 48:1073). Например, для определения гомологии, сходства или идентичности могут быть использованы BLAST или ClustalW от Национального Центра Биотехнологической Информации.
Гомологию, сходство или идентичность между полипептидами или полинуклеотидами можно определить путем сравнения информации о последовательностях с использованием, например, компьютерной программы GAP, такой как Needleman et at (1970), JMol Biol. 48:443, как раскрыто, например, в Smith and Waterman, Adv. Appl. Math (1981) 2:482. В общем, программа GAP определяет гомологию, сходство или идентичность в виде величины, получаемой путем деления количества выровненных одинаковых символов (т.е. нуклеотидов или аминокислот) на общее количество символов в более короткой из двух последовательностей. Параметры по умолчанию для программы GAP могут включать: (1) матрицу двоичного сравнения (содержащую величину 1 в случае идентичности и 0 в случае не идентичности) и взвешенную матрицу сравнения в соответствии с Gribskov et at (1986), Nucl. Acids Res. 14:6745, как раскрыто в Schwartz and Dayhoff, eds., Atlas of Protein Sequence and Structure, National Biomedical Research Foundation, pp.353-358 (1979) (или матрицу замен EDNAFULL (версия EMBOSS в NCBI NUC4.4)); (2) штраф 3,0 за каждый пропуск и дополнительно штраф 0,10 за каждый символ в каждом пропуске (или штраф за внесение пропуска 10 и штраф за удлинение пропуска 0,5) и (3) отсутствие штрафа за концевые пропуски.
В одном из воплощений вариант в соответствии с настоящей заявкой на изобретение может обладать активностью альдолазы aroG. В одном из воплощений вариант в соответствии с настоящей заявкой на изобретение может обладать активностью, способной увеличивать способность продуцировать аминокислоты с разветвленной цепью по сравнению с альдолазой дикого типа или не модифицированной альдолазой aroG. В одном из воплощений вариант в соответствии с настоящей заявкой на изобретение может обладать активностью, способной уменьшить уровень образования побочных продуктов в пути получения аминокислот с разветвленной цепью по сравнению с альдолазой дикого типа или не модифицированной альдолазой aroG. В одном из воплощений вариант в соответствии с настоящей заявкой на изобретение может обладать ослабленной активностью по сравнению с альдолазой дикого типа или не модифицированной альдолазой aroG, но не ограничиваться этим.
В еще одном аспекте настоящей заявки на изобретение предложен полинуклеотид, кодирующий вариант в соответствии с настоящей заявкой на изобретение.
Использованный здесь «полинуклеотид», который представляет собой полимер из нуклеотидов, состоящих из нуклеотидных мономеров, связанных в длинную цепь при помощи ковалентных связей, представляет собой цепь ДНК или РНК, имеющей по меньшей мере определенную длину. Конкретнее, он может относиться к полинуклеотидному фрагменту, кодирующему вариант.
Полинуклеотид, кодирующий вариант в соответствии с настоящей заявкой на изобретение, может включать нуклеотидную последовательность, кодирующую аминокислотную последовательность, представленную в SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 23 или SEQ ID NO: 25. В качестве примера настоящей заявки на изобретение полинуклеотид в соответствии с настоящей заявкой на изобретение может обладать или включать последовательность SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 24 или SEQ ID NO: 26. Кроме того, полинуклеотид в соответствии с настоящей заявкой на изобретение может состоять из или состоять по существу из последовательности SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 24 или SEQ ID NO: 26.
Полинуклеотид в соответствии с настоящей заявкой на изобретение может претерпевать различные модификации в кодирующей области в объеме, который не изменяет аминокислотную последовательность варианта в соответствии с настоящей заявкой на изобретение вследствие вырожденности кодонов или при рассмотрении кодонов, предпочтительных в организме, в котором вариант в соответствии с настоящей заявкой на изобретение должен экспрессироваться. В частности, полинуклеотид в соответствии с настоящей заявкой на изобретение может обладать или включать нуклеотидную последовательность, обладающую гомологией или идентичностью 70% или более, 75% или более, 80% или более, 85% или более, 90% или более, 95% или более, 96% или более, 97% или более, 98% или более и 100% или менее с последовательностью SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 24 или SEQ ID NO: 26, или может состоять из или состоять по существу из нуклеотидной последовательности, обладающей гомологией или идентичностью 70% или более, 75% или более, 80% или более, 85% или более, 90% или более, 95% или более, 96% или более, 97% или более, 98% или более и 100% или менее с последовательностью SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 24 или SEQ ID NO: 26, но не ограничиваться этим.
В частности, в последовательности, обладающей гомологией или идентичностью, кодоны, кодирующие аминокислоты, соответствующие положениям 217, 310, 403 и 462 в SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10, SEQ ID NO: 24 или SEQ ID NO: 26, могут представлять собой один из кодонов, кодирующих аланин.
Кроме того, полинуклеотид в соответствии с настоящей заявкой на изобретение может включать зонд, который может быть получен из известной генной последовательности, например, любой последовательности, которая может гибридизоваться с последовательностью, комплементарной всей или части полинуклеотидной последовательности в соответствии с заявкой на изобретение в строгих условиях. «Строгие условия» относятся условиям, при которых возможна специфическая гибридизация между полинуклеотидами. Такие условия, в частности, описаны в литературе (J. Sambrook et al., Molecular Clonong, A Laboratory Manual, 2nd Edition, Cold Spring Harbor Laboratory press, Cold Spring Harbor, New York, 1989; F.M. Ausubel et al., Current Protocols in Molecular Biology, John Wiley & Sons, Inc., New York, 9.50 9.51, 11.7 11.8). Например, строгие условия могут включать условия, при которых гены, обладающие высокой гомологией или идентичностью 70% или более, 75% или более, 80% или более, 85% или более, 90% или более, 95% или более, 96% или более, 97% или более, 98% или более или 99% или более, гибридизуются друг с другом, и гены, обладающие меньшей гомологией или идентичностью, чем вышеприведенные гомологии или идентичности, не гибридизуются друг с другом, или условия промывки при гибридизации по Саузерну, при которой промывку осуществляют однократно, в частности, два или три раза при концентрации соли и температуре, соответствующих 60°С, 1 × SSC (раствор цитрата и хлорида натрия), 0,1% SDS (додецилсульфат натрия), в частности, 60°С, 0,1 × SSC, 0,1% SDS, конкретнее, 68°С, 0,1 × SSC, 0,1% SDS.
Для гибридизации требуется, чтобы две нуклеиновые кислоты содержали комплементарные последовательности, хотя ошибки спаривания между основаниями возможны в зависимости от строгости условий гибридизации. Термин «комплементарный» используют для описания взаимодействия между нуклеотидными основаниями, которые могут гибридизоваться друг с другом. Например, в случае ДНК аденозин комплементарен тимину и цитозин комплементарен гуанину. Таким образом, полинуклеотид в соответствии с настоящей заявкой на изобретение может включать выделенные нуклеотидные фрагменты, комплементарные всей последовательности, а также последовательностям нуклеиновых кислот, по существу похожим на нее.
В частности, полинуклеотиды, обладающие гомологией или идентичностью с полинуклеотидом в соответствии с настоящей заявкой на изобретение, могут быть обнаружены с использованием условий гибридизации, включающих стадию гибридизации при величине Tm 55°С в вышеописанных условиях. Кроме того, величина Tm может составлять 60°С, 63°С или 65°С, но не ограничивается ими, и может быть подходящим образом скорректирована специалистом в данной области техники в зависимости от задачи.
Подходящая строгость условий для гибридизации полинуклеотидов зависит от длины полинуклеотидов и степени комплементарности, и эти переменные хорошо известны в области техники (например, Sambrook et al.).
В еще одном аспекте настоящей заявки на изобретение предложен вектор, содержащий полинуклеотид в соответствии с настоящей заявкой на изобретение. Этот вектор может представлять собой экспрессирующийся вектор для экспрессии полинуклеотида в клетке-хозяине, но не ограничиваться этим.
Используемый здесь термин «вектор» относится к конструкции ДНК, содержащей нуклеотидную последовательность полинуклеотида, кодирующего целевой полипептид, который функционально связан с подходящей областью для регулирования экспрессии (последовательностью, регулирующей экспрессию), таким образом, чтобы обеспечивать экспрессию целевого полипептида в подходящей клетке-хозяине. Последовательность, регулирующая экспрессию, может включать промотор, способный инициировать транскрипцию, любую операторную последовательность для регуляции такой транскрипции, последовательность, кодирующую подходящий сайт для связывания с рибосомой на мРНК, и последовательность, регулирующую прекращение транскрипции и трансляции. После трансформации в подходящую клетку-хозяина вектор может реплицироваться или функционировать независимо от генома хозяина, или сам может быть интегрирован в геном.
Вектор, используемый в настоящей заявке на изобретение не ограничен конкретным образом, и может быть использован любой вектор, известный в области техники. Примеры обычно используемого вектора могут включать природные или рекомбинантные плазмиды, космиды, вирусы и бактериофаги. Например, в качестве фагового вектора или космидного вектора могут быть использованы pWE15, М13, MBL3, MBL4, IXII, ASHII, APII, t10, t11, Charon4A и Charon21A и т.п., и в качестве плазмидного вектора могут быть использованы векторы на основе pDC, pBR, pUC, pBluescriptII, pGEM, pTZ, pCL, pET и т.п. В частности, могут быть использованы векторы pDC, pDCM2, pACYC177, pACYC184, pCL, pECCG117, pUC19, pBR322, pMW118 и pCC1BAC.
В одном из примеров полинуклеотид, кодирующий полипептид-мишень, может быть встроен в хромосому с использованием вектора для внутриклеточного встраивания хромосом. Встраивание полинуклеотида в хромосому может быть осуществлено без ограничения при помощи любого способа, известного в области техники, например, при помощи гомологичной рекомбинации. Вектор может дополнительно включать селективный маркер для подтверждения встраивания в хромосому. Селективный маркер предназначен для отбора клеток, трансформированных вектором, то есть для подтверждения того, что встроена целевая молекула нуклеиновой кислоты, и могут быть использованы маркеры, обеспечивающие селективные фенотипы, такие как резистентность к лекарственным средствам, потребность в питательных веществах, резистентность к цитотоксическим агентам и экспрессия поверхностных полипептидов. При обработке селективным агентом только клетки, экспрессирующие селективные маркеры, могут выжить или демонстрировать отличающиеся фенотипы в среде, и, таким образом, могут быть отобраны трансформированные клетки.
Используемый здесь термин «трансформация» относится к введению вектора, содержащего полинуклеотид, кодирующий целевой полипептид, в клетку-хозяина или микроорганизм таким образом, что полипептид, кодируемый полинуклеотидом, может экспрессироваться в данной клетке-хозяине. Пока трансформированный полинуклеотид может экспрессироваться в клетке-хозяине, не важно, интегрирован ли трансформированный полинуклеотид в хромосому клетки-хозяина и находится в ней, или расположен за пределами хромосомы, и оба случая могут быть включены. Кроме того, полинуклеотид может включать ДНК и РНК, кодирующие целевой полипептид. Полинуклеотид может быть введен в любой форме до тех пор, пока он может быть введен в клетку-хозяина и экспрессироваться в ней. Например, полинуклеотид может быть введен в клетку-хозяина в форме экспрессирующейся кассеты, которая представляет собой генетическую конструкцию, включающую все необходимые элементы, требующиеся для ее автономной экспрессии. Экспрессирующаяся кассета традиционно может включать промотор, функционально связанный с полинуклеотидом, сигнал прекращения транскрипции, сайт связывания с рибосомой и сигнал прекращения трансляции. Экспрессирующаяся кассета может находиться в форме экспрессирующегося вектора, способного к саморепликации. Кроме того, полинуклеотид без ограничения может быть введен в клетку-хозяина сам по себе и функционально связан с последовательностью, требующейся для его экспрессии в клетке-хозяине.
Кроме того, использованный здесь термин «функционально связанный» означает то, что последовательность полинуклеотида функционально связана с промоторной последовательностью, которая инициирует и опосредует транскрипцию полинуклеотида, кодирующего вариант-мишень в соответствии с настоящей заявкой на изобретения.
В еще одном аспекте настоящей заявки на изобретение предложен микроорганизм рода Corynebacterium, включающий один или более чем один из вариантов в соответствии с настоящей заявкой на изобретение, полинуклеотид в соответствии с настоящей заявкой на изобретение и вектор в соответствии с настоящей заявкой на изобретение.
Микроорганизм может включать вариант полипептида в соответствии с настоящей заявкой на изобретение, полинуклеотид, кодирующий этот полипептид, или вектор, содержащий полинуклеотид в соответствии с настоящей заявкой на изобретение.
Используемый здесь термин «микроорганизм» или «штамм» включает все микроорганизмы дикого типа или естественным образом или искусственно генетически модифицированные микроорганизмы, и он может представлять собой микроорганизм, в котором конкретный механизм ослаблен или усилен вследствие встраивания чужеродного гена или путем усиления или инактивации активности эндогенного гена, и может представлять собой микроорганизм, включающий генетическую модификацию, для продукции желаемого полипептида, белка или продукта.
Штамм в соответствии с настоящей заявкой на изобретение может представлять собой штамм, включающий любой один или более чем один из вариантов в соответствии с настоящей заявкой на изобретение, полинуклеотид в соответствии с настоящей заявкой на изобретение и вектор, содержащий полинуклеотид в соответствии с настоящей заявкой на изобретение; штамм, модифицированный для экспрессии варианта в соответствии с настоящей заявкой на изобретение или полинуклеотид в соответствии с настоящей заявкой на изобретение; штамм, экспрессирующий вариант в соответствии с настоящей заявкой на изобретение или полинуклеотид в соответствии с настоящей заявкой на изобретение (например, рекомбинантный штамм); или штамм, обладающий активностью варианта в соответствии с настоящей заявкой на изобретение (например, рекомбинантный штамм), но не ограничиваться этим.
Штамм в соответствии с настоящей заявкой на изобретение может представлять собой штамм, обладающий способностью продуцировать аминокислоты с разветвленной цепью.
Штамм в соответствии с настоящей заявкой на изобретение представляет собой микроорганизм, обладающий способностью в природе продуцировать альдолазу aroG или аминокислоты с разветвленной цепью, или микроорганизм, в котором вариант в соответствии с настоящей заявкой на изобретение или кодирующий его полинуклеотид (или вектор, содержащий полинуклеотид) встроен в альдолазу aroG, или родительский штамм, не обладающий способностью продуцировать аминокислоты с разветвленной цепью, и/или у которого способность продуцировать аминокислоту с разветвленной цепью нарушена, но не ограничивается этим.
В одном из примеров штамм в соответствии с настоящей заявкой на изобретение представляет собой клетку или микроорганизм, который трансформирован вектором, содержащим полинуклеотид в соответствии с настоящей заявкой на изобретение или полинуклеотид, кодирующий вариант в соответствии с настоящей заявкой на изобретение, для экспрессии варианта в соответствии с настоящей заявкой на изобретение, и для задачи настоящей заявки на изобретение штамм может включать все микроорганизмы, способные продуцировать аминокислоту с разветвленной цепью, включающие вариант в соответствии с настоящей заявкой на изобретение. Например, штамм в соответствии с настоящей заявкой на изобретение может представлять собой рекомбинантный штамм, в котором способность продуцировать аминокислоты с разветвленной цепью, увеличена путем экспрессии варианта альдолазы aroG за счет встраивания полинуклеотида, кодирующего вариант в соответствии с настоящей заявкой на изобретение в природном микроорганизме дикого типа или микроорганизме для продукции аминокислот с разветвленной цепью. Рекомбинантный штамм, в котором способность продуцировать аминокислоты с разветвленной цепью увеличена, может представлять собой микроорганизм, в котором способность продуцировать аминокислоты с разветвленной цепью увеличена по сравнению с природным микроорганизмом дикого типа или микроорганизмом с не модифицированной альдолазой (то есть, микроорганизмом, экспрессирующим альдолазу aroG дикого типа), но не ограничивается этим.
В одном из примеров микроорганизм с не модифицированной альдолазой aroG, представляющий собой штамм-мишень, сравниваемый для определения увеличения способности продуцировать аминокислоты с разветвленной цепью, может представлять собой штамм Corynebacterium glutamicum АТСС13032. В еще одном примере микроорганизм с не модифицированной альдолазой aroG, представляющий собой штамм-мишень, сравниваемый для определения увеличения способности продуцировать аминокислоты с разветвленной цепью, может представлять собой CJL-8109, KCCM12739P (СА10-3101) или KCCM11201P, но не ограничивается этим.
В одном из примеров рекомбинантный штамм может обладать увеличенной способностью продуцировать аминокислоты с разветвленной цепью приблизительно на 1% или более, в частности, приблизительно 3% или приблизительно 5%, по сравнению с родительским штаммом перед модификацией или не модифицированным микроорганизмом, но не ограничивается этим, до тех пор, пока он обладает увеличенной + величиной по сравнению с продуцирующей способностью родительского штамма до модификации или не модифицированным микроорганизмом.
В еще одном примере рекомбинантный штамм может обладать уменьшенной продукцией побочных продуктов, образующихся в пути продукции аминокислот с разветвленной цепью, приблизительно на 50% или менее, в частности, приблизительно 30% или менее или приблизительно 10% или менее по сравнению с родительским штаммом перед модификацией или не модифицированным микроорганизмом, но не ограничивается этим.
Используемый здесь термин «приблизительно» относится к диапазону, включающему все из ±0,5, ±0,4, ±0,3, ±0,2, ±0,1 и т.п., и он включает все величины, эквивалентные величинам, которые находятся немедленно после термина «более», или величинам в похожем диапазоне, но не ограничивается этим.
Использованный здесь термин «аминокислота с разветвленной цепью» относится к аминокислоте, имеющей разветвленную алкильную группу по боковой цепи, и включающей валин, лейцин и изолейцин. В частности, в настоящей заявке на изобретение аминокислота с разветвленной цепью может представлять собой аминокислоту с L-разветвленной цепью, и аминокислота с L-разветвленной цепью может представлять собой одну или более чем одну из L-валина, L-лейцина и L-изолейцина, но не ограничиваться этим.
В настоящей заявке на изобретение побочные продукты, образующиеся в пути продукции аминокислот с разветвленной цепью, относятся к веществам, отличающимся от аминокислот с разветвленной цепью, и, в частности, к ароматическим аминокислотам, и конкретнее, одной или более чем одной, выбранной из L-тирозина и L-фенилаланина, но не ограничиваться ими.
Используемый здесь термин «не модифицированный микроорганизм» не исключает штаммы, включающие мутацию, которая в природе может возникать у микроорганизмов, и может относиться к штамму дикого типа или самому природному штамму, или штамму до того, как характеристики меняются путем генетической модификации вследствие природных или искусственных факторов. Например, не модифицированный микроорганизм может относиться к штамму, в котором вариант альдолазы aroG в соответствии с настоящей заявкой на изобретение, описанный в описании настоящего изобретения, не встроен или к штамму до его встраивания. Термин «не модифицированный микроорганизм» может быть использован взаимозаменяемо с «штаммом перед модификацией», «микроорганизмом перед модификацией», «не мутантным штаммом», «не модифицированным штаммом», «не мутантным микроорганизмом» или «референсным микроорганизмом».
В одном из воплощений микроорганизм в соответствии с настоящей заявкой на изобретение может представлять собой Corynebacterium stationis, Corynebacterium crudilactis, Corynebacterium deserti, Corynebacterium efficiens, Corynebacterium callunae, Corynebacterium glutamicum, Corynebacterium singulare, Corynebacterium halotolerans, Corynebacterium striatum, Corynebacterium ammoniagenes, Corynebacterium pollutisoli, Corynebacterium imitans, Corynebacterium testudinoris или Corynebacterium flavescens.
Микроорганизм в соответствии с настоящей заявкой на изобретение может дополнительно включать модификацию для увеличения способности продуцировать аминокислоты с разветвленной цепью.
В одном из воплощений микроорганизм в соответствии с настоящей заявкой на изобретение может включать изменение активности одного или более чем одного из изопропилмалатсинтазы, гомосериндегидрогеназы, треониндегидратазы, аминотрансферазы аминокислот с разветвленной цепью и цитратсинтазы.
В одном из воплощений микроорганизм в соответствии с настоящей заявкой на изобретение может представлять собой микроорганизм, в котором активность одного или более чем одного из изопропилмалатсинтазы, аминотрансферазы аминкислот с разветвленной цепью, гомосериндегидрогеназы и треониндегидратазы дополнительно усилена.
В одном из воплощений микроорганизм в соответствии с настоящей заявкой на изобретение может представлять собой микроорганизм, в котором активность цитратсинтетазы дополнительно ослаблена.
Тем не менее, настоящая заявка на изобретение не ограничена вышеприведенным описанием, и специалист в данной области техники может подходящим образом выбрать дополнительные модификации, включенные в микроорганизм в соответствии с продуцируемыми аминокислотами с разветвленной цепью.
Используемое здесь «усиление» полипептидной активности означает то, что активность полипептида увеличивается по сравнению с собственной активностью. Усиление может быть использовано взаимозаменяемо с терминами, такими как активация, повышающая регуляция, сверхэкспрессия, увеличение и т.п. В частности, термины активация, усиление, повышающая регуляция, сверхэкспрессия и увеличение могут включать как случаи, при которых демонстрировалась активность, которая исходно не проявлялась, так и увеличенную активность по сравнению с собственной активностью или активностью до модификации. «Собственная активность» относится к активности конкретного полипептида, исходно проявляемой родительским штаммом или не модифицированным микроорганизмом до его трансформации, когда характеристики микроорганизма изменяются путем генетической модификации вследствие природных или искусственных факторов, и может быть использована взаимозаменяемо с «активностью до модификации». «Усиление», «повышающая регуляция», «сверхэкспрессия» или «увеличение» активности полипептида по сравнению с собственной активностью, означает то, что активность и/или концентрация (уровень экспрессии) улучшается по сравнению с активностью и/или концентрацией конкретного полипептида, исходно демонстрируемой родительским штаммом или немодифицированным микроорганизмом перед трансформацией.
Усиление может быть достигнуто путем встраивания чужеродного полипептида или путем усиления активности и/или увеличения концентрации (уровня экспрессии) эндогенного полипептида. Усиление активности полипептида можно подтвердить путем увеличения уровня активности полипептида, уровня экспрессии или количества продукта, высвободившегося из полипептида.
Усиление активности полипептида может быть достигнуто путем применения различных способов, хорошо известных в области техники, и эти способы не ограничены до тех пор, пока они могут усиливать активность полипептида-мишени по равнению с активностью микроорганизма перед модификацией. В частности, может быть использована генетическая инженерия и/или белковая инженерия, хорошо известные специалистам в данной области техники, которая представляет собой обычный способ молекулярной биологии, но способ не ограничен ими (например, Sitnicka et al. Functional Analysis of Genes. Advances in Cell Biology. 2010, Vol. 2. 1-16, Sambrook et al. Molecular Cloning 2012, и т.п.).
В частности, усиление активности полипептида в соответствии с настоящей заявкой на изобретение может быть достигнуто путем:
1) увеличения числа копий полинуклеотида, кодирующего полипептид, в клетках;
2) замены области, регулирующей экспрессию гена, кодирующего полипептид, в хромосоме на последовательность, обладающую сильной активностью;
3) модификации нуклеотидной последовательности, кодирующей стартовый кодон или 5'-UTR генного транскрипта, кодирующего полипептид;
4) модификации аминокислотной последовательности полипептида таким образом, что активности полипептида усиливается;
5) модификации полинуклеотидной последовательности, кодирующей полипептид, таким образом, что активность полипептида усиливается (например, модификация полинуклеотидной последовательности гена полипептида таким образом, что кодируется полипептид, модифицированный для усиления активности полипептида;
6) введения чужеродного полинуклеотида, обладающего активность полипептида или чужеродного кодирующего его полинуклеотида;
7) оптимизации кодона полинуклеотида, кодирующего полипептид;
8) анализа третичной структуры полипептида и, таким образом, выбора и модификации экспонируемого сайта, или химической модификации экспонируемого сайта; или
9) комбинации двух или более чем двух, выбранных из (1)-(8), но не ограничиваясь ими.
Конкретнее,
1) способ увеличения числа копий в клетке гена, колирующего полипептид, может быть достигнут путем введения вектора, который функционально связан с полинуклеотидом, кодирующим полипептид, и способен реплицироваться и функционировать независимо от клетки-хозяина в клетке-хозяине. В качестве альтернативы, этот способ может быть достигнут путем введения одной копии или двух копий полинуклеотидов, кодирующих полипептид, в хромосому клетки-хозяина. Введение в хромосому может быть осуществлено путем введения вектора, способного встраивать полинуклеотид в хромосому клетки-хозяина, в клетку-хозяина, но не ограничиваясь этим. Вектор является таким, как описано выше.
2) Способ замены области, регулирующей экспрессию (или последовательности, регулирующей экспрессию) гена, кодирующего полипептид, в хромосоме, на последовательность, обладающую сильной активностью, может быть достигнут путем индукции модификации последовательности путем делеции, вставки, не консервативной или консервативной замены, или путем их комбинации для дополнительного усиления активности области, регулирующей экспрессию, или путем замены последовательности на последовательность, обладающую более сильной активностью. Область, регулирующая экспрессию, может включать без ограничения промотор, операторную последовательность, последовательность, кодирующую сайт связывания с рибосомой, последовательность, регулирующую прекращение транскрипции или трансляции. В одном из примеров способ может включать замену исходного промотора на сильный промотор, но не ограничиваться этим.
Примеры сильного промотора могут включать промоторы cj1-cj7 (патент США № US 7662943 В2), промотор lac, промотор trp, промотор trc, промотор tac, промотор PR фага лямбда, промотор PL, промотор tet, промотор gapA, промотор SPL7, промотор SPL13(sm3) (патент США № US 10584338 В2), промотор 02 (патент США №US 10273491 В2), промотор tkt, промотор yccA, но не ограничиваться этим.
3) Способ модификации нуклеотидной последовательности, кодирующей стартовый кодон или 5'-UTR генного транскрипта, кодирующего полипептид, может быть достигнут, например, путем замены нуклеотидной последовательности на нуклеотидную последовательность, кодирующую другой стартовый кодон, обладающий более высоким уровнем экспрессии полипептида, чем эндогенный стартовый кодон, но не ограничивается этим.
4) и 5) Способ модификации аминокислотной последовательности или полинуклеотидной последовательности может быть достигнут путем индукции модификации последовательности путем делеции, вставки, не консервативной или консервативной замены аминокислотной последовательности полипептида или полинуклеотидной последовательности, кодирующей полипептид, или путем их комбинации для усиления активности полипептида, или путем замены последовательности на аминокислотную последовательность или полинуклеотидную последовательность, модифицированную таким образом, чтобы обладать более сильной активностью, или аминокислотную последовательность или полинуклеотидную последовательность, модифицированную для усиления активности. Замена в частности может быть осуществлена путем встраивания полинуклеотида в хромосому путем гомологичной рекомбинации, но не ограничиваясь этим. Используемый здесь вектор может дополнительно включать селектируемый маркер для подтверждения встраивания в хромосому. Селектируемый маркер является тем же самым, как описано выше.
6) Способ введения чужеродного полинуклеотида, обладающего активностью полипептида, может быть достигнут путем введения в клетку-хозяина чужеродного полинуклеотида, кодирующего полипептид, который демонстрирует активность, идентичную или похожую на активность полипептида. Чужеродный полинуклеотид может быть использован без ограничения независимо от его происхождения или последовательности при условии, что он демонстрирует активность, идентичную или похожую на активность полипептида. Введение может быть осуществлено специалистами в данной области техники путем подходящего выбора способа трансформации, известного в области техники, и экспрессия введенного полинуклеотида в клетке-хозяине обеспечивает получение полипептида, таким образом, увеличивая его активность.
7) Способ оптимизации кодона полинуклеотида, кодирующего полипептид, может быть достигнут путем оптимизации кодона эндогенного полинуклеотида для увеличения транскрипции или трансляции в клетке-хозяине, или путем оптимизации его кодонов таким образом, чтобы дать возможность достижения оптимизированной транскрипции и трансляции чужеродного полинуклеотида в клетке-хозяине.
8) Способ анализа третичной структуры полипептида и, таким образом, отбора и модификации экспонируемого сайта или его химической модификации может быть достигнут, например, путем сравнения информации о последовательности анализируемого полипептида с базой данных, в которой хранится информация о последовательностях известных белков, для определения кандидатов белков-матриц в соответствии со степенью сходства последовательности, и, таким образом, подтверждения структуры, основанной на информации, таким образом, отбора и трансформации или модификации экспонируемого сайта, который предполагается модифицировать или химически модифицировать.
Такое усиление полипептидной активности может означать то, что активность или концентрация (уровень экспрессии) полипептида увеличена по сравнению с активностью или концентрацией полипептида, экспрессируемого в штамме дикого типа или микробном штамме до модификации, или то, что количество продукта, продуцируемого из полипептида, увеличено, но не ограничивается этим.
Модификация всего полинуклеотида или его части в микроорганизме в соответствии с настоящей заявкой на изобретение может быть достигнута путем (а) гомологичной рекомбинации с использованием вектора для встраивания хромосомы в микроорганизм или редактирования генома с использованием сконструированной нуклеазы (например, CRISPR-Cas9) и/или (б) может быть вызвана светом, таким как ультрафиолетовые лучи и излучение и т.п., и/или химическими веществами, но не ограничивается этим. Способ модификации части или всего гена может включать способ с использованием технологии рекомбинантной ДНК. Например, путем введения нуклеотидной последовательности или вектора, содержащего нуклеотидную последовательность, гомологичную гену-мишени, в микроорганизм для того, чтобы вызвать гомологичную рекомбинацию, может быть удален весь ген или его часть. Введенная нуклеотидная последовательность или вектор может включать доминирующий селективный маркер, но не ограничиваться этим.
Используемый здесь термин «ослабление» полипептидной активности является всесторонним понятием, включающим как уменьшенную активность, так и отсутствие активности по сравнению с его собственной активностью. Ослабление может быть использовано взаимозаменяемо с такими терминами, как инактивация, дефицит, понижающая регуляция, уменьшение, сокращение, затухание и т.п.
Ослабление может также включать случай, когда сама полипептидная активность уменьшена или отсутствует по сравнению с активностью полипептида, исходно демонстрируемой микроорганизмом вследствие мутации полинуклеотида, кодирующего полипептид; случай, когда общий уровень внутриклеточной полипептидной активности и/или концентрации (уровень экспрессии) уменьшается по сравнению с природным штаммом вследствие ингибирования экспрессии гена полинуклеотида, кодирующего полипептид, или ингибирования трансляции в полипептид и т.п.; случай, когда полинуклеотид вообще не экспрессируется; и/или случай, когда не обнаруживается никакая полипептидная активностью даже при экспрессии полинуклеотида. Используемый здесь термин «собственная активность» относится к активности конкретного полипептида, исходно находившегося в родительском штамме перед трансформацией, микроорганизме дикого типа или не модифицированном микроорганизме, когда характеристика изменена вследствие генетической модификации, вызванной природным или искусственным фактором, и может быть использован взаимозаменяемо с «активностью перед модификацией». Выражение «полипептидная активность «инактивирована, устранена, уменьшена, подвергнута понижающей регуляции, снижена или ослаблена» по сравнению с его собственной активностью» означает то, что полипептидная активностью уменьшена по сравнению с активностью конкретного полипептида, исходно демонстрируемой родительским штаммом до трансформации или не модифицированным микроорганизмом.
Ослабление активности полипептида может быть осуществлено при помощи любого способа, известного в области техники, но способ не ограничен этим, и может быть достигнуто путем применения различных способов, известных в области техники (например Nakashima N et al., Bacterial cellular engineering by genome editing and gene silencing, Int J Mol Sci. 2014; 15(2):2773-2793, Sambrook et al. Molecular Cloning 2012, и т.п.).
В частности, ослабление полипептида в соответствии с настоящей заявкой на изобретение может быть достигнуто путем:
1) удаления части или всего гена, кодирующего полипептид;
2) модификации области, регулирующей экспрессию (последовательности, регулирующей экспрессию), таким образом, что экспрессия гена, кодирующего полипептид, уменьшается;
3) модификации аминокислотной последовательности, составляющей полипептид, таким образом, что полипептидная активность устраняется или ослабляется (например, делеция/замена/вставка одной или более чем одной аминокислоты в аминокислотную последовательность).
4) модификации генной последовательности, кодирующей полипептид, таким образом, что полипептидная активность устраняется или ослабляется (например, делеция/замена/вставка одного или более чем одного нуклеотида в нуклеотидной последовательности гена полипептида для кодирования полипептида, который модифицирован для удаления или ослабления активности полипептида;
5) модификации нуклеотидной последовательности, кодирующей инициирующий кодон или 5'-UTR генного транскрипта, кодирующего полипептид;
6) введения антисмыслового олигонуклеотида (например, антисмысловой РНК), который комплементарно связывается с транскриптом гена, кодирующего полипептид;
7) добавления последовательности, комплементарной последовательности Шайна-Дальгарно (SD) на переднем конце последовательности SD гена, кодирующего полипептид, с образованием вторичной структуры, таким образом, ингибируя прикрепление рибосомы;
8) инжиниринга путем обратной транскрипции (RTE), который добавляет промотор, который должен быть подвергнут обратной транскрипции, по 3' концу открытой рамки считывания (ORF) генной последовательности, кодирующей полипептид; или
9) комбинации двух или более чем двух, выбранных из способов (1)-(8) выше, но не ограничиваясь этим.
Например,
1) Способ удаления части или всего гена, кодирующего полипептид, может быть осуществлен путем удаления всего полинуклеотида, кодирующего эндогенный целевой полипептид в хромосоме, или путем замены полинуклеотида на полинуклеотид или маркерный ген, имеющий частично удаленную последовательность нуклеиновой кислоты.
Кроме того, 2) способ модификации экспрессии регулирующей области (экспрессия регулирующей последовательности) может быть осуществлен путем индукции модификации в области, регулирующей экспрессию (последовательности, регулирующей экспрессию) путем делеции, вставки, не консервативной замены или консервативной замены, или их комбинации; или путем замены последовательности на последовательность, обладающую более слабой активностью. Область, регулирующая экспрессию, может включать промотор, операторную последовательность, последовательность, кодирующую сайт связывания с рибосомой, и последовательность для регуляции транскрипции и трансляции, но не ограничена ими.
Кроме того, 5) способ модификации нуклеотидной последовательности, кодирующей инициирующий кодон или 5'-UTR генного транскрипта, кодирующего полипептид, может быть осуществлен, например, путем замены нуклеотидной последовательности на нуклеотидную последовательность, кодирующую другой инициирующий кодон, обладающий более низким уровнем экспрессии полипептида, чем эндогенный инициирующий кодон, но не ограничивается этим.
Кроме того, 3) и 4) способ модификации аминокислотной последовательности или полинуклеотидной последовательности может быть осуществлен путем индукции модификации последовательности путем делеции, вставки, не консервативной или консервативной замены аминокислотной последовательности полипептида или полинуклеотидной последовательности, кодирующей полипептид, или их комбинации для дополнительного ослабления активности полипептида, или путем замены последовательности на аминокислотную последовательность или полинуклеотидную последовательность, модифицированную таким образом, чтобы обладать более слабой активностью, или аминокислотную последовательность или полинуклеотидную последовательность, модифицированную таким образом, чтобы не обладать активностью. Например, экспрессия гена может быть подавлена или ослаблена путем введения в полинуклеотидную последовательность мутации таким образом, чтобы образовывать стоп-кодон, но не ограничивается этим.
6) Способ введения антисмыслового олигонуклеотида (например, антисмысловой РНК), который комплементарно связывается с транскриптом гена, кодирующего полипептид, можно найти в литературе [Weintraub, Н. et al., Antisense-RNA as a molecular tool for genetic analysis, Reviews - Trends in Genetics, Vol. 1(1) 1986].
7) Способ добавления последовательности, комплементарной последовательности Шайна-Дальгарно (SD) относительно переднего конца последовательности SD гена, кодирующего полипептид, с образованием вторичной структуры, таким образом, подавление присоединения рибосомы может быть достигнуто путем подавления трансляции мРНК или уменьшения его скорости.
8) Инжиниринг путем обратной транскрипции (RTE), который добавляет промотор, который должен быть подвергнут обратной транскрипции по 3'-концу открытой рамки считывания (ORF) генной последовательности, кодирующей полипептид, может быть осуществлен путем формирования антисмыслового нуклеотида, комплементарного генному транскрипту, кодирующему полипептид, для ослабления активности.
В микроорганизме в соответствии с настоящей заявкой на изобретение варианты, полинуклеотиды, векторы и аминокислоты с разветвленной цепью являются такими же как описанные в других аспектах.
В еще одном аспекте настоящей заявки на изобретение предложен способ получения аминокислот с разветвленной цепью, включающий культивирование в среде микроорганизма рода Corynebacterium.
Использованный здесь термин «культивирование» означает то, что микроорганизм рода Corynebacterium в соответствии с настоящей заявкой на изобретение выращивают в надлежащим образом контролируемых условиях окружающей среды. Процесс культивирования в соответствии с настоящей заявкой на изобретение может быть осуществлен в подходящей культуральной среде и условиях культивирования, известных в области техники. Такой процесс культивирования может быть легко скорректирован для применения специалистом в данной области техники в соответствии с выбранным штаммом. В частности, культивирование может представлять собой периодическое культивирование, непрерывное культивирование и/или культивирование с подпиткой, но не ограничиваться этим.
Использованный здесь термин «среда» относится к смеси веществ, которая содержит в качестве основного ингредиента питательные вещества, требующиеся для культивирования микроорганизма рода Corynebacterium в соответствии с настоящей заявкой на изобретение, и она обеспечивает питательные вещества и факторы роста, а также воду, которые необходимы для жизнедеятельности и роста. В частности, среда и другие условия культивирования, используемые для выращивания микроорганизма рода Corynebacterium в соответствии с настоящей заявкой на изобретение, могут представлять собой любую среду, используемую для обычного культивирования микроорганизмов без какого-либо конкретного ограничения. Тем не менее, микроорганизм рода Corynebacterium в соответствии с настоящей заявкой на изобретение может быть культивирован в аэробных условиях в обычной среде, содержащей подходящий источник углерода, источник азота, источник фосфора, неорганическое соединение, аминокислоту и/или витамин, при корректировании температуры, рН и т.п.
В частности, состав среды для культивирования микроорганизма рода Corynebacterium можно найти в литературе ["Manual of Methods for General Bacteriology" by the American Society for Bacteriology (Washington D.C., USA, 1981)].
В настоящей заявке на изобретение источник углерода может включать углеводы, такие как глюкоза, сахароза, лактоза, фруктоза, сахароза, мальтоза и т.п.; сахарные спирты, такие как маннит, сорбит и т.п.; органические кислоты, такие как пировиноградная кислота, молочная кислота, лимонная кислота и т.п.; аминокислоты, такие как глутаминовая кислота, метионин, лизин и т.п. Кроме того, источник углерода может включать природные органические питательные вещества, такие как гидролизат крахмала, мелассы, сырые мелассы, рисовые отруби, маниок, выжимки сахарного тростника, жидкий кукурузный экстракт и т.п. В частности, могут быть использованы углеводы, такие как глюкоза и стерилизованные предварительно обработанные мелассы (т.е. мелассы, преобразованные в восстанавливающие сахара), и, кроме того, различные другие источники углерода в подходящем количестве могут быть использованы без ограничения. Эти источники углерода могут быть использованы сами по себе или в комбинации двух или более чем двух из них, но не ограничиваться этим.
Источник азота может включать источники неорганического азота, такие как аммиак, сульфат аммония, хлорид аммония, ацетат аммония, фосфат аммония, карбонат аммония, нитрат аммония и т.п.; аминокислоты, такие как глутаминовая кислота, метионин, глутамин и т.п.; и источники органического азота, такие как пептон, NZ-амин, мясной экстракт, дрожжевой экстракт, солодовый экстракт, жидкий кукурузный экстракт, гидролизат казеина, рыба или продукты ее деградации, обезжиренный соевый жмых или продукт его деградации и т.п. Эти источники азота могут быть использованы сами по себе или в комбинации двух или более чем двух из них, но не ограничиваться этим.
Источник фосфора может включать первичный кислый фосфат калия, вторичный кислый фосфат калия или соответствующие содержащие натрий соли и т.п. Примеры неорганического соединения могут включать хлорид натрия, хлорид кальция, хлорид железа, сульфат магния, сульфат железа, сульфат марганца, карбонат кальция и т.п. Кроме того, могут быть включены аминокислоты, витамины и/или соответствующие предшественники. Эти составляющие ингредиенты или предшественники могут быть добавлены в среду партиями или непрерывным образом, но эти источники фосфора не ограничиваются этим.
рН среды может быть скорректирован во время культивирования микроорганизма рода Corynebacterium путем добавления в среду подходящим образом соединения, такого как гидроксид аммония, гидроксид калия, аммиак, фосфорная кислота, серная кислота и т.п. Кроме того, в процессе культивирования для предотвращения образования пены может быть добавлен пеногаситель, такой как сложный эфир полигликоля и жирной кислоты. Кроме того, кислород или содержащий кислород газ могут быть введены в среду для поддержания в среде аэробного состояния; или азот, водород или газообразный диоксид углерода могут быть введены без нагнетания в нее газа для поддержания анаэробного или микро аэробно го состояния среды, но газ не ограничен ими.
Температура среды для культивирования в соответствии с настоящей заявкой на изобретение может находиться в диапазоне от 20°С до 45°С, и, в частности, от 25°С до 40°С, и культивирование можно осуществлять в течение приблизительно от 10 до 160 часов, но не ограничиваться этим.
Аминокислота с разветвленной цепью, получаемая в результате культивирования в соответствии с настоящей заявкой на изобретение, может быть высвобождена в среду или оставаться в клетках.
Способ получения аминокислоты с разветвленной цепью в соответствии с настоящей заявкой на изобретение дополнительно может включать стадию приготовления микроорганизма рода Corynebacterium в соответствии с настоящей заявкой на изобретение, стадию приготовления среды для культивирования штамма или их комбинацию (независимо от последовательности, в любой последовательности), например, до стадии культивирования.
Способ получения аминокислот с разветвленной цепью в соответствии с настоящей заявкой на изобретение дополнительно может включать стадию выделения аминокислоты с разветвленной цепью из культуральной среды (среды, в которой осуществлялось культивирование) или микроорганизма рода Corynebacterium в соответствии с настоящей заявкой на изобретение. Стадия выделения может быть дополнительно включена после стадии культивирования.
На стадии выделения желаемая аминокислота с разветвленной цепью может быть собрана с использованием способа культивирования микроорганизма в соответствии с настоящей заявкой на изобретение, например, с использованием подходящего способа, известного в области техники, в соответствии с периодическим способом культивирования, непрерывным способом культивирования или культивированием с подпиткой. Например, могут быть использованы способы, такие как центрифугирование, фильтрование, обработка агентом, осаждающим белок (способ высаливания), экстракция, ультразвуковое разрушение, ультрафильтрация, диализ, различные виды хроматографий, такие как хроматография на молекулярных ситах (гель-фильтрация), адсорбционная хроматография, ионообменная хроматография, аффинная хроматография и т.п., HPLC (высокоэффективная жидкостная хроматография) или их комбинация, и желаемая аминокислота с разветвленной цепью может быть выделена из среды или микроорганизмов с использованием подходящих способов, известных в области техники.
Кроме того, способ получения аминокислот с разветвленной цепью в соответствии с заявкой на изобретение дополнительно может включать процесс очистки, который может быть осуществлен с использованием подходящего способа, известного в области техники. В одном из примеров, когда способ получения аминокислот с разветвленной цепью в соответствии с настоящей заявкой на изобретение включает как стадию выделения, так и стадию очистки, тогда стадия выделения и стадия очистки могут быть осуществлены непрерывно или с перерывами независимо от последовательности, или могут быть осуществлены периодически (или непрерывно) независимо от последовательности или одновременно, или могут быть объединены в одну стадию, но способ не ограничивается этим.
В способе в соответствии с настоящей заявкой на изобретение варианты, полинуклеотиды, векторы и микроорганизмы являются такими, как описано в других аспектах.
В еще одном аспекте настоящей заявки на изобретение предложена композиция, для получения аминокислот с разветвленной цепью, включающая вариант в соответствии с настоящей заявкой на изобретение, полинуклеотид, кодирующий вариант, вектор, содержащий полинуклеотид, или микроорганизм рода Corynebacterium, включающий полинуклеотид в соответствии с настоящей заявкой на изобретение; содержащую его среду; или комбинацию двух или более чем двух из них.
Композиция в соответствии с настоящей заявкой на изобретение может дополнительно включать любой подходящий эксципиент, обычно используемый в композиции для получения аминокислот с разветвленной цепью, и такой эксципиент может представлять собой, например, консервант, увлажнитель, диспергирующий агент, суспендирующий агент, буферный агент, стабилизатор или изотонический агент, но не ограничиваться этим.
В композиции в соответствии с настоящей заявкой на изобретение варианты, полинуклеотиды, векторы, штаммы, среды и аминокислоты с разветвленной цепью являются такими, как описано в других аспектах.
В еще одном аспекте настоящей заявки на изобретение предложено применение получения аминокислоты с разветвленной цепью вариантом альдолазы aroG в соответствии с настоящей заявкой на изобретение.
В соответствии с еще одним аспектом настоящей заявки на изобретение предложено применение получения аминокислоты с разветвленной цепью микроорганизмом, включающим одно или более чем одно, выбранное из вариантов альдолазы aroG в соответствии с настоящей заявкой на изобретение; полинуклеотида, кодирующего вариант альдолазы aroG; и вектора, содержащего полинуклеотид.
При применении настоящей заявки на изобретение варианты, полинуклеотиды, векторы, микроорганизмы и т.п. являются такими, как описано в других аспектах.
Способ осуществления изобретения
Настоящая заявка на изобретение будет описана подробно при помощи примеров. Тем не менее, специалистам в данной области техники понятно то, что эти примеры приведены исключительно для задач иллюстрации, и не предназначены для ограничения объема изобретения.
Пример 1: Обнаружение мутации альдолазы aroG
Пример 1-1. Получение вектора, содержащего альдолазу aroG
Для получения библиотеки мутантов альдолазы aroG, обладающих активностью фосфо-2-дегидро-3-дезоксигептаноат альдолазы, сначала готовили рекомбинантный вектор, содержащий альдолазу aroG. Для амплификации гена aroG (SEQ ID NO: 2), кодирующего альдолазу aroG (SEQ ID NO: 1, KEGG ID: NCgl2098), полученного из Corynebacterium glutamicum дикого типа осуществляли ПЦР на основе хромосомы дикого типа Corynebacterium glutamicum АТСС13032 в качестве матрицы с использованием праймеров SEQ ID NO: 11 и 12, в следующих условиях: 25 циклов денатурации при 94°С в течение 1 минуты, отжига при 58°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты с использованием ДНК-полимеразы Pfu. Конкретные последовательности используемых праймеров представлены в таблице 1. Амплифицированный продукт клонировали в вектор pCR2.1 Е. coli с использованием набора для клонирования ТОРО Cloning Kit (Invitrogen) с получением «pCR-aroG».

Пример 1-2. Получение библиотеки мутантов альдолазы aroG
На основе вектора, полученного в примере 1-1, библиотеку мутаций альдолазы aroG получали с использованием набора для ПЦР сниженной точности (clontech Diversify® PCR Random Mutagenesis Kit). Реакцию ПЦР осуществляли с использованием SEQ ID NO: 11 и SEQ ID NO: 12 в качестве праймеров в условиях, при которых могут возникать от 0 до 3 мутаций на 1000 п.о. (пар оснований). В частности, после предварительного нагревания при 94°С в течение 30 секунд осуществляли ПЦР путем повторения 25 циклов денатурации при 94°С в течение 30 секунд и отжига при 68°С в течение 1 минуты и 30 секунд. С использованием полученного таким образом продукта ПЦР в качестве мегапраймера (от 50 до 125 нг) осуществляли ПЦР путем повторения 25 циклов денатурации при 95°С в течение 50 секунд, отжига при 60°С в течение 50 секунд и полимеризации при 68°С в течение 12 минут, и получающиеся в результате продукты обрабатывали DpnI, трансформировали в Е. coli DH5a при помощи способа теплового шока и высевали на твердую среду LB (Луриа-Бертани), содержащую канамицин (25 мг/л). 20 видов трансформированных таким образом колоний отбирали с получением плазмид, и нуклеотидные последовательности анализировали. В результате подтвердили то, что мутации были введены в различные позиции с частотой 2 мутации/кб. Приблизительно 20000 трансформированных колоний Е. coli собирали и плазмиды выделяли, и они были названы «рТОРО-aroG-library».
Пример 2: Оценка полученной библиотеки и отбор вариантов
Пример 2-1. Отбор мутантных штаммов, обладающих увеличенной способностью продуцировать L-лейцин
pTOPO-aroG-library, полученную в примере 1-2, трансформировали в Corynebacterium glutamicum АТСС13032 дикого типа путем электропорации, и затем высевали на питательную среду (таблица 2), содержащую 25 мг/л канамицина для отбора 10000 колоний штаммов, в которые встроен мутантный ген. Каждая отобранная колония была названа от АТСС13032/рТОРО_aroG(mt)1 до ATCC13032/pTOPO_aroG(mt) 10000.
Ферментационный титр оценивали следующим образом для каждой колонии для идентификации колоний среди 10000 полученных колоний, в которых продукция L-лейцина увеличивалась, а L-тирозина и L-фенилаланина среди ароматических аминокислот уменьшалась.


Платиновую петлю с каждой колонией инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мкг/мл канамицина в 25 мл автоклавированной среды для продуцирования (таблица 2), и затем культивировали при 30°С в течение 60 часов при встряхивании при 200 об./мин. После завершения выращивания продукцию L-лейцина и L-тирозина, и L-фенилаланина среди ароматических аминокислот измеряли посредством высокоэффективной жидкостной хроматографии (HPLC, SHIMAZDU LC20A).
Среди полученных таким образом 10000 колоний отбирали четыре штамма с наиболее улучшенной способностью продуцировать L-лейцин по сравнению со штаммом Corynebacterium glutamicum (АТСС13032) дикого типа, которые были названы АТСС13032/pTOPO_aroG(mt)2256, ATCC13032/pTOPO_aroG(mt)6531, АТСС13032/pTOPO_aroG(mt)8316 и ATCC13032/pTOPO_aroG(mt)9426. Концентрации L-лейцина (Leu), L-тирозина (Tyr) и L-фенилаланина (Phe), полученные в выбранных штаммах, представлены в таблице 3 ниже.

В соответствии с представленным в таблице 3 выше было подтверждено, что Corynebacterium glutamicum ATCC13032/pTOPO_aroG(mt)2256, имеющий мутацию в гене aroG, продемонстрировал увеличенную способность продуцировать L-лейцин приблизительно в 1,4-раза по сравнению с Corynebacterium glutamicum АТСС13032, представляющим собой родительский штамм. Кроме того, подтвердили то, что ATCC13032/pTOPO_aroG(mt)6531, АТСС 13032/pTOPO_aroG(mt)8316, АТСС13032/рТОРО aroG(mt)9426 продемонстрировал улучшенную способность продуцировать L-лейцин приблизительно в 1,4-раза по сравнению с родительским штаммом. Кроме того, подтвердили то, что все четыре штамма ATCC13032/pTOPO_aroG(mt)2256, ATCC13032/pTOPO_aroG(mt)6531, АТСС13032/рТОРО aroG(mt)8316, АТСС13032/рТОРО aroG(mt)9426 продемонстрировали уменьшение продукции L-тирозина в 2,4-8,5-раз и продукции L-фенилаланина в 3-10-раз.
Пример 2-2. Подтверждение мутаций в мутантных штаммах с увеличенной продукцией L-лейцина и уменьшенной продукцией ароматических аминокислот
Для подтверждения мутации в гене aroG в четырех выбранных мутантных штаммах осуществляли ПЦР с использованием праймеров с SEQ ID NO: 11 и SEQ ID NO: 12, перечисленных в таблице 1, на основе ДНК каждого мутантного штамма в условиях денатурации при 94°С в течение 5 минут с последующими 30 циклами денатурации при 94°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд, и затем полимеризации при 72°С в течение 5 минут, и затем осуществляли секвенирование ДНК.
В качестве результата секвенирования подтвердили то, что в штамме АТСС13032/рТОРО aroG (mt)2256, CGC, 649-й, 650-й и 651-й нуклеотиды гена aroG, заменены на GCG. Последнее свидетельствует о том, что может кодироваться вариант (далее, R217A) в котором аргинин, который представляет собой 217-ю аминокислоту альдолазы aroG, заменен на аланин. Аминокислотная последовательность мутанта альдолазы aroG (R217A) и нуклеотидная последовательность мутанта альдолазы aroG, кодирующая его, представлены в SEQ ID NO: 3 и 4.
Кроме того, в штамме АТСС 13032/pTOPO_aroG(mt)6531 подтвердили то, что АА, 928-й и 929-й нуклеотиды гена aroG, заменены на GC. Последнее указывает на то, что может кодироваться вариант (далее, К310А), в котором лизин, представляющий собой 310-ю аминокислоту альдолазы aroG, заменен на аланин. Аминокислотная последовательность мутанта альдолазы aroG (К310А) и нуклеотидная последовательность мутанта альдолазы aroG, кодирующая его, представлены в SEQ ID NO: 5 и 6.
В штамме ATCC13032/pTOPO_aroG(mt)8316 подтвердили то, что CGC, которые представляют собой с 1207-го по 1209-й нуклеотиды гена aroG, заменены на GCG. Последнее указывает на то, что может кодироваться вариант (далее, R403A), в котором аргинин, представляющий собой 403-ю аминокислоту альдолазы aroG, заменен на аланин. Аминокислотная последовательность мутанта альдолазы aroG (R403A) и нуклеотидная последовательность мутанта альдолазы aroG, кодирующая его, представлены в SEQ ID NO: 7 и 8.
В штамме АТСС13032/рТОРО aroG(mt)9426 подтвердили то, что АА, 1385-й и 1386-й нуклеотиды гена DAHP, заменены на CG. Последнее свидетельствует о том, что может кодироваться мутант (далее, Е462А), в котором глутамат, 462-я аминокислота альдолазы aroQ заменена на аланин. Аминокислотная последовательность мутанта альдолазы aroG (Е462А) и нуклеотидная последовательность мутанта альдолазы aroG, кодирующая его, представлены в SEQ ID NO: 9 и 10.
Таким образом, в следующих примерах эксперименты осуществляли для того, чтобы определить то, влияют ли мутации (R217A, K310A, R403A, Е462А) на продукцию L-лейцина и ароматических аминокислот в микроорганизмах рода Corynebacterium.
Пример 3: Подтверждение способности продуцировать L-лейцин, L-тирозин, и L-фенилаланин выбранными мутантными штаммами
Пример 3-1. Получение встраиваемых векторов, содержащих мутацию альдолазы aroG
Для встраивания мутаций, выбранных в примере 2, в штаммы, получали встраиваемые векторы. Сайт-направленный мутагенез использовали для получения векторов для встраивания мутации aroG (R217A, K310A, R403A, Е462А). Осуществляли ПЦР с использованием пар праймеров SEQ ID NO: 13 и 14 и SEQ ID NO: 15 и 16 для получения мутации R217A, и с использованием пар праймеров SEQ ID NO: 13 и 17 и SEQ ID NO: 15-18 для получения мутации K310A, основанной на хромосомах дикого типа Corynebacterium glutamicum АТСС13032 в качестве матрицы. Кроме того, осуществляли ПЦР с использованием пар праймеров SEQ ID NO: 13 и 19 и SEQ ID NO: 15 и 20 для получения мутации R403A, и с использованием пар праймеров SEQ ID NO: 13 и 21 и SEQ ID NO: 15-22 для получения мутации Е462А. В частности, осуществляли ПЦР в условиях денатурации при 94°С в течение 5 минут с последующими 30 циклами денатурации при 94°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд и затем полимеризации при 72°С в течение 5 минут. Конкретные последовательности используемых праймеров представлены в таблице 4.


Получающиеся в результате продукты ПЦР клонировали путем слияния гомологичной последовательности 15-ти концевых оснований между фрагментами ДНК с использованием линейного вектора pDCM2 (Публикация заявки на Корейский патент KR 10-2020-0136813 А), расщепленного ферментом рестрикции SmaI, и фермента для слияния с получением векторов для замены аминокислот, а именно, «PDCM2-aroG(R217A)», «pDCM2-aroG (K310A)», «pDCM2-aroG(R403A)» и «pDCM2-aroG(E462A)». Кроме того, векторы для замены аминокислот в положениях 217, 310, 403 и 462 в aroG, «pDCM2-aroG (R217A, K310A, Е462А)» и «pDCM2-aroG (R217A, K310A, R403A, Е462А)», получали путем комбинирования вариантов. Аминокислотная последовательность вариантов альдолазы aroG (R217A, K310A, Е462А) и нуклеотидная последовательность вариантов альдолазы aroG, кодирующая их, представлены в SEQ ID NO: 23 и 24. Кроме того, аминокислотные последовательности вариантов альдолазы aroG (R217A, K310A, R403A, Е462А) и нуклеотидные последовательности вариантов альдолазы aroG, кодирующие их, представлены в SEQ ID NO: 25 и 26.
Пример 3-2. Введение вариантов в штамм АТСС13032 и их оценка
Векторы pDCM2-aroG (R217A), pDCM2-aroG (К310А), pDCM2-aroG (R403A), pDCM2-aroG (E462A), pDCM2-aroG (R217A, K310A, E462A) и pDCM2-aroG (R217A, K310A, R403A, E462A), полученные в примере 3-1, трансформировали в АТСС 13032 путем электропорации, и штаммы, в которых были встроены векторы в хромосому путем рекомбинации гомологичной последовательности, отбирали в среде, содержащей 25 мг/л канамицина. Отобранные первичные штаммы вновь подвергали вторичному кроссоверу, и отбирали штаммы, в которые была введена мутация целевого гена. Введение мутации гена aroG в окончательные трансформированные штаммы подтверждали путем осуществления ПЦР с использованием праймеров с SEQ ID NO: 11 и SEQ ID NO: 12, перечисленных в таблице 1, и последующего анализа нуклеотидных последовательностей. В общей сложности получали 5 штаммов, которые были названы ATCC13032_aroG_R217A, ATCC13032_aroG_K310A, ATCC13032_aroG_R403A, ATCC13032_aroG_E462A, ATCC13032_aroG_(R217A, K310A, E462A) и ATCC13032 aroG (R217A, K310A, R403A, E462A).
Ферментационный титр в колбе определяли для оценки способности продуцировать L-лейцин и ароматические аминокислоты всеми 6 штаммами, полученными выше. Одну платиновую петлю каждого полученного выше родительского штамма Corynebacterium glutamicum АТСС 13032, и АТСС13032 aroG R217A, АТСС13032 aroG К310А, АТСС13032 aroG R403A, ATCC13032_aroG_E462A, ATCC13032_aroG_(R217A, K310A, E462A), и ATCC13032 aroG (R217A, K310A, R403A, E462A) инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мл среды для продуцирования, и затем культивировали при встряхивании при 200 об/мин при 30°С в течение 60 часов для получения L-лейцина. После завершения культивирования продукцию L-лейцина, L-тирозина и L-фенилаланина измеряли при помощи HPLC. Концентрация лейцина в культуральной среде для каждого тестируемого штамма представлена в таблице 5 ниже.

В соответствии с представленным в таблице 5 выше подтвердили то, что ATCC13032_aroG_R217A, АТСС 13032_aroG_K310A, АТСС 13032_aroG_R403A, ATCC13032_aroG_E462A, ATCC13032_aroG_(R217A, K310A, Е462А), и АТСС13032 aroG (R217A, K310A, R403A, Е462А) продемонстрировали улучшенный выход L-лейцина приблизительно в 1,35 раз 1,55 раз по сравнению с родительским штаммом Corynebacterium glutamicum АТСС 13032. Кроме того, подтвердили то, что выход L-тирозина уменьшился в 2,5 - 8,5 раз, и выход L-фенилаланина уменьшился приблизительно в 3,5-14 раз.
Пример 4: Подтверждение способности продуцировать лейцин, тирозин и фенилаланин в отобранном штамме с мутациями aroG, продуцирующем лейцин
Даже если штамм дикого типа рода Corynebacterium обладает способностью продуцировать лейцин, получают только весьма следовое количество. Соответственно, эксперимент осуществляли для получения лейцин-продуцирующего штамма, полученного из АТСС 13032, и для введения выбранных мутаций для подтверждения способности продуцировать лейцин, тирозин и фенилаланин. Конкретный эксперимент был проведен следующим образом.
Пример 4-1. Получение штамма CJL-8109, продуцирующего L-лейцин
В качестве штамма для продукции L-лейцина с высокой концентрацей получали штамм, являющийся производным АТСС 13032, содержащий: (1) мутацию (R558H), при которой G, 1673-й нуклеотид гена leuA, заменен на А, и аргинин, представляющий собой 558-ю аминокислоту белка LeuA, заменен на гистидин; (2) мутацию (G561D), при которой GC, 1682-й и 1683-й нуклеотиды гена leuA, заменены на AT, и глицин, представляющий собой 561-ю аминокислоту, заменен на аспарагиновую кислоту; и (3) мутацию (Р247С), при которой СС, 739-й и 740-й нуклеотиды гена leuA, заменены на TG, и пролин, представляющий собой 247-ю аминокислоту, заменен на цистеин.
В частности, вектор pDCM2-leuA(P247C, R558H, G561D), содержащий мутации гена leuA, трансформировали в Corynebacterium glutamicum АТСС 13032 путем электропорации, и штамм, в котором вектор был встроен в хромосому путем рекомбинации гомологичной последовательности, был отобран в среде, содержащей 25 мг/л канамицина. Отобранный первичный штамм вновь подвергали вторичному кроссоверу, и отбирали штамм, в который были введены мутации гена leuA. Введение мутации в окончательно трансформированный штамм подтверждали путем осуществления ПЦР с использованием праймеров с SEQ ID NO: 27 и SEQ ID NO: 28 в соответствии с таблицей 6 в условиях денатурации при 94°С в течение 5 минут с последующими 30 циклами денатурации при 94°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд, и затем полимеризации при 72°С в течение 5 минут и анализа нуклеотидных последовательностей. Штамм ATCC13032_leuA_(P247C, R558H, G561D), трансформированый вектором pDCM2-leuA(P247C, R558H, G561D), был назван «CJL-8105».


Для увеличения способности продуцировать L-лейцин в полученном таким образом штамме CJL-8105, получали штамм, в который вводили мутант ilvE (V156A) гена, кодирующего аминотрансферазу аминокислоты с разветвленной цепью (Корейский патент №10-2143964). В частности, вектор pDCM2-ilvE(V156A), содержащий мутацию гена ilvE, трансформировали в Corynebacterium glutamicum CJL-8105 путем электропорации, и штамм, в который вектор был встроен в хромосому путем рекомбинации гомологичной последовательности, был отобран в среде, содержащей 25 мг/л канамицина. Отобранные первичные штаммы вновь подвергали вторичному кроссоверу, и отбирали штамм, в который была введена мутация гена ilvE. Введение мутации в окончательно трансформированный штамм подтверждали путем осуществления ПЦР с использованием праймеров с SEQ ID NO: 29 и SEQ ID NO: 30 в соответствии с таблицей 7 в условиях денатурации при 94°С в течение 5 минут с последующими 30 циклами денатурации при 94°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд, и затем полимеризации при 72°С в течение 5 минут, и анализа нуклеотидных последовательностей, таким образом, подтверждая введение мутации V156A. Штамм, трансформированный вектором pDCM2-ilvE(V156A), был назван «CJL-8108».

Для увеличения способности продуцировать L-лейцин в полученном таким образом штамме CJL-8108 получали штамм с введенным мутантом gltA (M312I; SEQ ID NO: 41), обладающий ослабленной активностью цитратеинтазы. В частности, вектор pDCM2-gltA(M3121), содержащий мутацию гена gltA, трансформировали в Corynebacterium glutamicum CJL-8108 путем электропорации, и штамм, в котором вектор был введен в хромосому путем рекомбинации гомологичной последовательности, был отобран в среде, содержащей 25 мг/л канамицина. Отобранный первичный штамм вновь подвергали вторичному кроссоверу, и отбирали штамм, в который была введена мутация гена gltA. Введение мутации в окончательно трансформированный штамм подтверждали путем осуществления ПЦР с использованием праймеров с SEQ ID NO: 31 и SEQ ID NO: 32 в соответствии с таблицей 8 в условиях денатурации при 94°С в течение 5 минут, с последующими 30 циклами денатурации при 94°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд, и затем полимеризации при 72°С в течение 5 минут, и анализа нуклеотидных последовательностей, таким образом, подтверждая введение мутации M312I. Штамм, трансформированный вектором pDCM2-gltA(M3121), был назван «CJL-8109».

Пример 4-2. Введение варианта альдолазы aroG в штамм CJL-8109 и его оценка
CJL-8109, который представляет собой штамм, продуцирующий лейцин, трансформировали векторами pDCM2-aroG(R217A), pDCM2-aroG(K310A), pDCM2-aroG(R403A), PDCM2-aroG(E462A), pDCM2-aroG(R217A, K310A, E462A) и pDCM2-aroG(R217A, K310A, R403A, E462A), полученными в примере 3-1, и штаммы, в которых векторы были встроены в хромосому путем рекомбинации гомологичной последовательности, отбирали в среде, содержащей 25 мг/л канамицина. Отобранные первичные штаммы вновь подвергали вторичному кроссоверу, и отбирали штаммы, в которые была введена мутация гена-мишени. Введение мутации гена aroG в окончательно трансформированный штамм подтверждали путем осуществления ПЦР с использованием праймеров с SEQ ID NO: 11 и SEQ ID NO: 12 и анализа нуклеотидных последовательностей, таким образом, подтверждая введение мутаций альдолазы aroG в штаммы. Полученный CJL-8109_aroG_R217A был назван CJL-8110, CJL-8109_aroG_K310A был назван CJL-8111, CJL-8109_aroG_R403A был назван CJL-8112, CJL-8109 aroG Е462А был назван CJL-8113, CJL-8109 aroG (R217A, К310А, Е462А) был назван СА13-8114 и CJL-8109 aroG (R217A, K310A, R403A, Е462А) был назван CJL-8115.
Оценивали способность продуцировать лейцин полученными таким образом штаммами CJL-8110, CJL-8111, CJL-8112, CJL-8113, СА13-8114, CJL-8115 и АТСС13032, CJL-8109. Культивирование в колбе осуществляли тем же самым образом, как в примере 2, и продукцию лейцина и ароматических аминокислот измеряли при помощи HPLC после завершения культивирования, и результаты культивирования представлены в таблице 9 ниже.
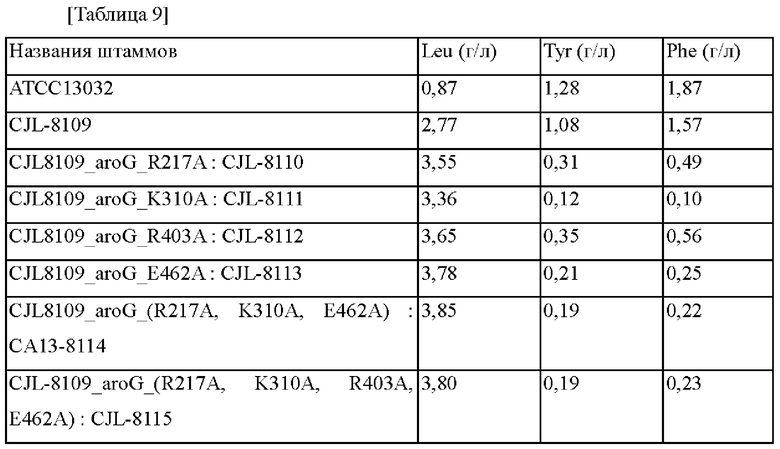
В соответствии с представленным в таблице 9, штаммы Corynebacterium glutamicum CJL-8110, CJL-8111, CJL-8112, CJL-8113, CA13-8114 и CJL-8115, продуцирующие L-лейцин, дополнительно имеющие мутации R217A, K310A, R403A и Е462А в гене aroG, продемонстрировали увеличенную продукцию лейцина приблизительно в 4-5 раз по сравнению с родительским штаммом Corynebacterium glutamicum АТСС13032. Кроме того, штаммы Corynebacterium glutamicum CJL-8110, CJL-8111, CJL-8112, CJL-8113, CA13-8114 и CJL-8115, продуцирующие L-лейцин, продемонстрировали улучшенную способность продуцировать L-лейцин приблизительно в 1,2-1,6 раз, и уменьшенную способность продуцировать L-тирозин и способность продуцировать L-фенилаланин приблизительно в 10-20 раз по сравнению с родительским штаммом Corynebacterium glutamicum CJL-8109. В соответствии с вышеприведенными результатами подтвердили то, что аминокислоты в положениях 217, 310, 403 и 462 в аминокислотной последовательности альдолазы aroG представляют собой важные положения для продукции L-лейцина, и L-тирозина и L-фенилаланина, представляющих собой ароматические аминокислоты.
Полученный таким образом штамм СА13-8114 был депонирован в Корейском центре культур микроорганизмов (KCCM), представляющем собой Международный депозитарий, в соответствии с Будапештским договором о международном признании депонирования микроорганизмов для целей патентной процедуры 18 января 2021 года под №КССМ12931Р.
Пример 5. Подтверждение способности продуцировать лейцин, тирозин и фенилаланин в отобранном штамме с мутацией aroG, продуцирующем изолейцин
Для подтверждения того, оказывают ли отобранные мутации действие в отношении изолейцина, который представляет собой типичную аминокислоту с разветвленной цепью, как лейцин, был проведен эксперимент для подтверждения способности продуцировать изолейцин путем введения мутации в штамм, продуцирующий изолейцин, рода Corynebacterium. Конкретные эксперименты проводили следующим образом.
Пример 5-1. Получение штамма, продуцирующего L-изолейцин СА10-3101 Штамм, продуцирующий L-изолейцин, разработали из Corynebacterium glutamicum АТСС13032 дикого типа. В частности, для высвобождения обратного ингибирования треонина, представляющего собой предшественник изолейцина в пути биосинтеза, аргинин, представляющий собой 407-ю аминокислоту в hom, ген, кодирующий гомосериндегидрогеназу, заменен на гистидин (US 2020-0340022 Al). В частности, для получения штаммов, в которые введен hom(R407H), осуществляли ПЦР с использованием пар праймеров с SEQ ID NO: 33 и 34 или SEQ ID NO: 35 и 36, основанных на хромосоме Corynebacterium glutamicum АТСС 13032, в качестве матрицы. Последовательности используемых здесь праймеров представлены в таблице 10 ниже.
PfuUltra™, представляющую собой полимеразу ДНК с высокой степенью надежности (Stratagene), использовали в качестве полимеразы для реакции ПЦР, и осуществляли ПЦР путем повторения 28 циклов денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты. В результате получали каждый из фрагментов ДНК (1000 п.о.) в области 5' против хода транскрипции и фрагмент ДНК (1000 п.о.) в области 3' по ходу транскрипции вокруг мутации гена hom. ПЦР осуществляли на основе двух амплифицированных фрагментов ДНК в качестве матрицы с использованием праймеров с SEQ ID NO: 34 и SEQ ID NO: 35 в условиях денатурации при 95°С в течение 5 минут, с последующими 28 циклами денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 2 минут, и затем полимеризации при 72°С в течение 5 минут.
В результате амплифицировали фрагмент ДНК (2 кб), включающий мутацию гена hom, кодирующего вариант гомосериндегидрогеназы, при которой аргинин в положении 407 заменен на гистидин. Амплифицируемый продукт очищали с использованием набора для очистки ПЦР (QIAGEN) и использовали в качестве встраиваемого фрагмента ДНК для получения вектора. В то же самое время, после обработки очищенного амплифицированного продукта ферментом рестрикции SmaI, отношение мольной концентрации (М) вектора pDCM2, подвергнутого термической обработке при 65°С в течение 20 минут для встраивания фрагмента ДНК, амплифицируемого продукта, устанавливали в отношении 1:2, и вектор клонировали с использованием Набора для клонирования путем слияния (TaKaRa) в соответствии с инструкцией от производителя для получения таким образом вектора pDCM2-R407H для встраивания мутации hom(R407H) в хромосому.
Полученный таким образом вектор трансформировали в Corynebacterium glutamicum АТСС13032 путем электропорации и подвергали вторичному кроссоверу с получением штамма, включающего мутацию hom(R407H) в хромосоме, и штамм был назван Corynebacterium glutamicum АТСС13032 hom(R407H).
Для высвобождения обратного действия в отношении L-изолейцина и увеличения активности в полученном АТСС13032 hom(R407H) получали штамм, в который введен мутант ilvA (Т381А, F383A), который представляет собой ген, кодирующий L-треониндегидратазу. Конкретнее, для получения штаммов, в которые введены мутации ilvA(T381A, F383A), осуществляли ПЦР с использованием пар праймеров с SEQ ID NO: 37 и 38 или SEQ ID NO: 39 и 40, основанных на хромосоме Corynebacterium glutamicum АТСС 13032, в качестве матрицы. Последовательности используемых здесь праймеров представлены в таблице 11 ниже.


PfuUltra™, представляющую собой полимеразу ДНК с высокой степенью надежности (Stratagene), использовали в качестве полимеразы для реакции ПЦР, и осуществляли ПЦР путем повторения 28 циклов денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты. В качестве результата получали каждый из фрагмента ДНК (1126 п. о.) в области 5' против хода транскрипции и фрагмент ДНК (286 п. о.) в области 3' по ходу транскрипции вокруг мутации гена ilvA. ПЦР осуществляли на основе двух амплифицированных фрагментов ДНК в качестве матрицы с использованием праймеров с SEQ ID NO: 37 и SEQ ID NO: 40 в условиях денатурации при 95°С в течение 5 минут, с последующими 28 циклами денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 2 минут, и затем полимеризации при 72°С в течение 5 минут.
В результате амплифицировали фрагмент ДНК (1,4 кб), включающий мутацию гена ilvA, кодирующий вариант треониндегидрогеназы, при которой треонин в положении 381 заменен на аланин и фенилаланин в положении 383 заменен на аланин. Амплифицируемый продукт очищали с использованием набора для очистки ПЦР (QIAGEN) и использовали в качестве встраиваемого фрагмента ДНК для получения вектора. В то же самое время, после обработки очищенного амплифицированного продукта ферментом рестрикции SmaI, отношение мольной концентрации (М) вектора pDCM2, подвергнутого термической обработке при 65°С в течение 20 минут для встраивания фрагмента ДНК, амплифицируемого продукта, устанавливали в отношении 1:2, и вектор клонировали с использованием Набора для клонирования путем слияния (TaKaRa) в соответствии с инструкцией от производителя для получения таким образом вектора pDCM2-ilvA(T381A, F383A) для встраивания мутации ilvA(T381A, F383A) в хромосому. Полученный таким образом вектор трансформировали в Corynebacterium glutamicum АТСС 13032 hom(R407H) путем электропорации и подвергали вторичному кроссоверу с получением штамма, включающему мутацию ilvA(T381A, F383A) на хромосоме, и штамм был назван Corynebacterium glutamicum СА10-3101.
Пример 5-2. Введение вариантов альдолазы aroG в штамм СА10-3101 и их оценка
СА10-3101, который представляет собой штамм, продуцирующий L-изолейцин, трансформировали векторами pDCM2-aroG (R217A), pDCM2-aroG(K310A), pDCM2-aroG(R403A), pDCM2-aroG(E462A), pDCM2-aroG(R217A, K310A, E462A) и pDCM2-aroG(R217A, K310A, R403A, E462A), полученными в примере 3-1, и штаммы, в которых векторы были встроены в хромосому путем рекомбинации гомологичной последовательности, отбирали в среде, содержащей 25 мг/л канамицина. Отобранные первичные штаммы вновь подвергали вторичному кроссоверу, и отбирали штаммы, в которые вводили мутацию гена-мишени. Введение мутации гена aroG в окончательно трансформированные штаммы подтверждали путем осуществления ПЦР с использованием праймеров с SEQ ID NO: 11 и SEQ ID NO: 12 и анализа нуклеотидных последовательностей, таким образом, подтверждая введение в штаммы мутации альдолазы aroG.
Полученные таким образом штаммы были названы СА10-3101 aroG R217A, CA10-3101_aroG_K310A, CA10-3101_aroG_R403A, CA10-3101_aroG_E462A, CA10-3101_aroG_(R217A, K310A, E462A), и CA10-3101_aroG_(R217A, K310A, R403A, E462A).
Оценивали способность продуцировать L-изолейцин полученными штаммами CA10-3101_aroG_R217A, CA10-3101_aroG_K310A, CA10-3101_aroG_R403A, CA10-3101_aroG_E462A, CA10-3101_aroG_(R217A, K310 A, E462A), CA10-3101 aroG (R217A, K310A, R403A, E462A) и ATCC13032, CA10-3101. L-Изолейцин получали путем инокулирования родительского штамма и штаммов с мутацией альдолазы aroG в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мл среды для продуцирования изолейцина, и затем культивирования при встряхивании при 32°С в течение 60 часов при 200 об./мин.
Композиция среды для продуцирования, используемая в этом примере, является следующей.
<Среда для продуцирования>
10% Глюкоза, 0,2% дрожжевой экстракт, 1,6% (NH4)2SO4, 0,1% KH2PO4, 0,1% MgSO4⋅7H2O, 10 мг/л FeSO4⋅7H2O, 10 мг/л MnSO4⋅H2O, 200 мкг/л биотина, рН 7,2
После завершения культивирования продукцию L-изолейцина и побочных продуктов измеряли с использованием высокоэффективной жидкостной хроматографии (HPLC), и концентрации L-изолейцина и побочных продуктов в культуральной среде для каждого тестируемого штамма представлены в таблице 12 ниже.

В соответствии с представленным в таблице 12 штаммы, продуцирующие L-изолейцин, имеющие дополнительные мутации R217A, K310A, R403A и Е462А в гене aroG, продемонстрировали увеличенную способность продуцировать L-изолейцин приблизительно в 1,1-1,2 раза по сравнению с родительским штаммом Corynebacterium glutamicum СА10-3101, и уменьшенную способность продуцировать L-тирозин и способность продуцировать L-фенилаланин.
В соответствии с вышеприведенными результатами можно подтвердить то, что аминокислоты в положениях 217, 310, 403 и 462 в аминокислотной последовательности альдолазы aroG представляют собой важные положения для продукции L-изолейцина и L-тирозина, и L-фенилаланина, которые представляют собой ароматические аминокислоты.
Пример 6: Подтверждение способности продуцировать валил в отобранном штамме с мутацией aroG, продуцирующем валин
Для подтверждения того, оказывают ли отобранные мутации действие в отношении L-валина, который представляет собой типичную аминокислоту с разветвленной цепью, как лейцин, отобранные мутации также вводили в штамм, продуцирующий валин, KCCM11201P рода Corynebacterium для подтверждения способности продуцировать валин. Конкретные эксперименты осуществляли следующим образом.
Пример 6-1. Введение вариантов альдолазы aroG в штамм KCCM11201P и их оценка
Для подтверждения того, являются ли мутации эффективными для увеличения способности продуцировать валин, использовали Corynebacterium glutamicum KCCM11201P (US 8465962 В2), представляющий собой штамм, продуцирующий L-валин. KCCM11201P, который представляет собой штамм, продуцирующий валин, трансформировали векторами pDCM2-aroG (R217A), pDCM2-aroG(K310A), pDCM2-aroG(R403A), pDCM2-aroG(E462A), pDCM2-aroG(R217A, K310A, E462A) и pDCM2-aroG(R217A, K310A, R403A, E462A), полученными в примере 3-1, и штаммы, в которых векторы были встроены в хромосому путем рекомбинации гомологичной последовательности, отбирали в среде, содержащей 25 мг/л канамицина. Отобранные первичные штаммы вновь подвергали вторичному кроссоверу, и отбирали штаммы, в которые была введена мутация гена-мишени. Введение мутации гена aroG в окончательно трансформированные штаммы подтверждали путем осуществления ПЦР с использованием праймеров с SEQ ID NO: 11 и SEQ ID NO: 12 и анализа нуклеотидных последовательностей, таким образом, подтверждая введение в штаммы мутации альдолазы aroG. Полученные таким образом штаммы были названы KCCM11201P - aroG(R217A), KCCM11201P - aroG(K310A), KCCM11201P - aroG(R403A), KCCM11201P - aroG(E462A), KCCM11201P - aroG(R217A, K310A, E462A) и KCCM11201P - aroG(R217A, K310A, R403A, E462A).
Оценивали способность продуцировать валин полученными штаммами KCCM11201P - aroG(R217A), KCCM11201P - aroG(K310A), KCCM11201P - aroG (R403A), KCCM11201P - aroG (E462A), KCCM11201P - aroG(R217A, K310A, E462A) и KCCM11201P - aroG(R217A, K310A, R403A, E462A). Культивирование в колбе осуществляли тем же самым образом, как в примере 2, и продукцию валина измеряли при помощи HPLC после завершения культивирования, и результаты культивирования представлены в таблице 13 ниже.
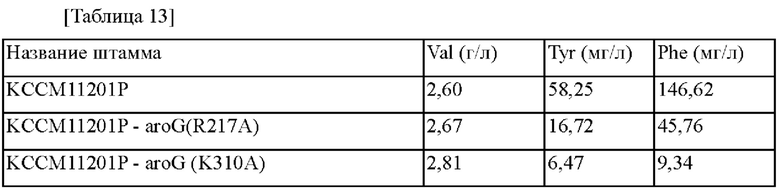

В соответствии с представленным в таблице 13 выше штаммы Corynebacterium glutamicum, продуцирующие L-валин, КССМ11201Р - aroG (R217A), KCCM11201P - aroG (K310A), KCCM11201P - aroG (R403A), KCCM11201P - aroG (E462A), KCCM11201P - aroG(R217A, K310A, E462A), и KCCM11201P - aroG (R217A, K310A, R403A, E462A), имеющие дополнительные мутации R217A, K310A, R403A и Е462А в гене aroG, продемонстрировали увеличенную способность продуцировать валин максимально в 1,11 раз и уменьшенную способность продуцировать L-тирозин и способность продуцировать L-фенилаланин приблизительно в 10-20 раз по сравнению с родительским штаммом Corynebacterium glutamicum KCCM11201P.
В соответствии с вышеприведенными результатами можно подтвердить то, что аминокислоты в положениях 217, 310, 403 и 462 в аминокислотной последовательности альдолазы aroG представляют собой важные положения для продукции L-валина и L-тирозина, и L-фенилаланина, которые представляют собой ароматические аминокислоты.
Референсный пример 1: Подтверждение продукции лейцина вследствие мутации gltA(M3121)
Референсный пример 1-1. Получение встраиваемого вектора, содержащего мутацию gltA
Сайт-направленный мутагенез использовали для получения вектора для встраивания мутации gltA (M312I; SEQ ID NO: 41).
Осуществляли ПЦР с использованием пар праймеров с SEQ ID NO: 43 и 44 и SEQ ID NO: 45 и 46, основанных на хромосомах Corynebacterium glutamicum дикого типа. Осуществляли ПЦР в условиях денатурации при 94°С в течение 5 минут с последующими 30 циклами денатурации при 94°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд, и затем полимеризации при 72°С в течение 5 минут. Получающиеся в результате генные фрагменты клонировали путем слияния гомологичной последовательности 15-ти концевых оснований между фрагментами ДНК с использованием линейного вектора pDCM2, расщепленного ферментом рестрикции SmaI, и фермента для слияния для получения вектора pDCM2-gltA(M3121) для замены метионина, который представляет собой 312-ю аминокислоту, на изолейцин.

Референсный пример 1-2. Введение вариантов в штамм АТСС13032 и их оценка
Вектор pDCM2-gltA(M3121) трансформировали в АТСС13032 дикого типа, и штамм, в который вектор был встроен в хромосому путем рекомбинации гомологичной последовательности, был отобран в среде, содержащей 25 мг/л канамицина. Отобранный первичный штамм вновь подвергали вторичному кроссоверу, и отбирали штамм, в который была введена мутация целевого гена. Введение мутации гена gltA в окончательно трансформированный штамм подтверждали путем осуществления ПЦР с использованием праймеров с SEQ ID NO: 31 и SEQ ID NO: 32 (пример 4-1, таблица 8) и анализа нуклеотидных последовательностей, таким образом, подтверждая введение мутации (SEQ ID NO: 42) в штамм. Полученный таким образом штамм был назван АТСС13032 gltA M312I.
Ферментационный титр в колбе определяли для оценки способности продуцировать лейцин полученным выше штаммом АТСС 13032_gltA_M312I. Одну платиновую петлю каждого полученного выше родительского штамма Corynebacterium glutamicum АТСС13032 и АТСС13032 gltA M312I инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мл среды для продуцирования, и затем выращивали при встряхивании при 200 об/мин при 30°С в течение 60 часов для получения лейцина. После завершения культивирования продукцию лейцина измеряли при помощи HPLC. Концентрация лейцина в культуральной среде для каждого тестируемого штамма представлена в таблице 15 ниже.
- Среда для продуцирования: 100 г глюкозы, 40 г (NH4)2SO4, 2,5 г соевого белка, 5 г сухого кукурузного экстракта, 3 г мочевины, 1 г KH2PO4, 0,5 г MgSO4⋅7H2O, 100 мкг биотина, 1000 мкг тиамина гидрохлорида, 2000 мкг пантотената кальция, 3000 мкг никотинамида, 30 г СаСО3 (на основе 1 л дистиллированной воды), рН 7,0

На основе этих результатов подтвердили то, что замена M312I в gltA представляет собой эффективную мутацию для увеличения продукции лейцина.
Референсный пример 2: Подтверждение продукции изолейцина вследствие мутации ilvA(T381A, F383A)
Референсный пример 2-1: Получение pECCG117-ilvA(F383A)
Для амплификации ilvA (SEQ ID NO: 48), гена, кодирующего треониндегидратазу (SEQ ID NO: 47), сайты для фермента рестрикции BamHI вводили по обоим концам праймеров (SEQ ID NO: 49 и SEQ ID NO: 50) для амплификации от области промотора (приблизительно 300 п.о. против хода транскрипции относительно стартового кодона) до терминаторной области (приблизительно 100 п.о. по ходу транскрипции относительно стартового кодона) на основе последовательности ilvA, о которой ранее сообщалось, в которую введена мутация F383A (World J Microbiol Biotechnol (2015) 31:1369-1377). Кроме того, праймеры (SEQ ID NO: 51 и SEQ ID NO: 52) использовали для встраивания мутации F383A в ilvA. Последовательности используемых здесь праймеров представлены в таблице 16 ниже.

Осуществляли ПЦР с использованием пар праймеров с SEQ ID NO: 49 и 52 и SEQ ID NO: 50 и 51 на основе хромосомы дикого типа Corynebacterium glutamicum АТСС13032. ПЦР осуществляли в условиях денатурации при 95°С в течение 5 минут с последующими 30 циклами денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд и затем полимеризации при 72°С в течение 5 минут.
В результате фрагмент ДНК (1460 п.о.) в области 5' против хода транскрипции и фрагмент ДНК (276 п.о.) в области 3' по ходу транскрипции получали вокруг мутации гена ilvA.
ПЦР осуществляли на основе двух амплифицированных фрагментов ДНК в качестве матрицы с использованием праймеров с SEQ ID NO: 49 и SEQ ID NO: 50.
В результате амплифицировали фрагмент ДНК (1531 п.о.), включающий мутацию гена ilvA, при которой 383-й фенилаланин заменен на аланин. Вектор pECCG117 (Корейский патент №10-0057684) и фрагмент ДНК ilvA, обработанный ферментом рестрикции BamHI, лигировали с использованием лигазы ДНК и клонировали с получением плазмиды, и плазмида была названа pECCG117-ilvA(F383A).
Референсный пример 2-2: Дополнительное введение произвольной мутации в pECCG117 ilvA(F383A)
Для получения варианта гена, кодирующего L-треониндегидратазу, получали плазмиду с мутантным геном ilvA с использованием набора для произвольного мутагенеза (Agilent Technologies, USA). ПЦР осуществляли с использованием праймеров с SEQ ID NO: 49 и SEQ ID NO: 50, основанных на хромосомах, ilvA(F383A) в качестве матрицы. ПЦР осуществляли в условиях денатурации при 95°С в течение 2 минут с последующими 30 циклами денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд, и затем полимеризации при 72°С в течение 10 минут.
В качестве результата амплифицировали фрагмент ДНК (1531 п.о.), который представляет собой вариант ilvA, способный кодировать L-треониндегидратазу, имеющий произвольную мутацию дополнительно к мутации, при которой 383-й фенилаланин заменен на аланин. Вектор pECCG117 и фрагмент ДНК мутанта ilvA обрабатывали ферментом рестрикции BamHI, лигировали с использованием лигазы ДНК и клонировали с получением группы плазмид.
Референсный пример 2-3: Получение штамма CJILE-301
pECCG117-ilvA(F383A) вводили в штамм Corynebacterium glutamicum АТСС13032 hom(R407H), и штамм, в который была введена полученная в результате плазмида, был назван hom(R407H)/pECCG117-ilvA(F383A). Кроме того, группу вариантов плазмид, полученных в референсном примере 2-2, вводили в штамм Corynebacterium glutamicum АТСС 13032 hom(R407H) и высевали на минимальную среду, и определяли уровень гибели. В качестве результата уровень гибели достигал 70%, и жизнеспособные клетки инокулировали в среду для посева и культивировали. Наконец, был отобран мутантный штамм, демонстрирующий превосходную способность продуцировать изолейцин по сравнению с контролем, АТСС 13032 hom(R407H)/pECCG117-ilvA(F383A), и назван Corynebacterium glutamicum CJLE-301.
В результате секвенирования гена ilvA путем выделения плазмиды из штамма CJILE-301 подтвердили то, что штамм может кодировать мутантный белок, в котором 381-й Т заменен на А, дополнительно к мутации, при которой А, представляющий собой 1141-й нуклеотид гена ilvA, заменен на G, приводя к мутации, при которой 383-й F белка ilvA заменен на А, и он представлен в SEQ ID NO: 54.
Референсный пример 2-4: Введение вариантов ilvA (Т381А, F383A)
Праймеры с SEQ ID NO: 49 и SEQ ID NO: 52 (пример 5-1, таблица 16) получали для встраивания вариантов ilvA (T381A, F383A) в штамм дикого типа.
Для получения штамма, в который вводили варианты ilvA (Т381А, F383A), осуществляли ПЦР с использованием праймеров с SEQ ID NO: 49 и SEQ ID NO: 52 на основе плазмидной ДНК, экстрагированной из штамма CJILE-301 в качестве матрицы.
PfuUltra™, представляющую собой полимеразу ДНК с высокой степенью надежности (Stratagene), использовали в качестве полимеразы для реакции ПЦР, и осуществляли ПЦР путем повторения 28 циклов денатурации при 95°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 2 минут.
В качестве результата получали фрагмент гена (1411 п.о.), включающий терминаторную область приблизительно 100 п.о. гена ilvA (1311 п.о.).
Амплифицируемый продукт очищали с использованием набора для очистки ПЦР (QIAGEN) и использовали в качестве встраиваемого фрагмента ДНК для получения вектора. В то же самое время, после обработки очищенного амплифицированного продукта ферментом рестрикции SmaI отношение мольной концентрации (М) вектора pDCM2, подвергнутого термической обработке при 65°С в течение 20 минут для встраивания фрагмента ДНК, амплифицируемый продукт устанавливали в отношении 1:2 и вектор клонировали с использованием набора для клонирования путем слияния (TaKaRa) в соответствии с инструкцией от производителя для получения, таким образом, вектора pDCM2-T381A_F383A для встраивания мутаций Т381А и F383A в хромосому.
Полученный таким образом вектор трансформировали в Corynebacterium glutamicum АТСС 13032 hom(R407H) путем электропорации и подвергали вторичному кроссоверу, таким образом, получая штамм, включающий мутацию ilvA(T381A, F383A; SEQ ID NO: 53) на хромосоме, и штамм был назван СА10-3101.
Штамм СА10-3101 был депонирован в Корейском центре культур микроорганизмов (KCCM), представляющий собой Международный депонирующий орган, в соответствии с Будапештским договором о международном признании депонирования микроорганизмов для целей патентной процедуры 27 мая 2020 года под № KCCM12739P.
Штамм KCCM 12739Р инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мл среды для продуцирования изолейцина, и затем культивировали при встряхивании при 32°С в течение 60 часов при 200 об/мин для получения L-изолейцина. Композиция используемой среды для продуцирования была следующей.
<Среда для продуцирования>
10% глюкоза, 0,2% дрожжевой экстракт, 1,6% (NH4)2SO4, 0,1% KH2PO4, 0,1% MgSO4⋅7H2O, 10 мг/л FeSO4⋅7H2O, 10 мг/л MnSO4⋅H2O, 200 мкг/л биотина, рН 7,2
После завершения культивироавния концентрации L-изолейцина и L-треонина в культуральной среде измеряли с использованием высокоэффективной жидкостной хроматографии (HPLC), и результаты представлены в таблице 17 ниже.

В соответствии с представленным в таблице 17 родительский штамм Corynebacterium glutamicum АТСС 13032 hom(R407H) не обладал способностью продуцировать L-изолейцин, но мутантный штамм АТСС13032 hom(R407H) ilvA(T381A, F383A) был способен продуцировать L-изолейцин с концентрацией 3,9 г/л, таким образом, подтверждая то, что продуктивность в отношении L-изолейцина значительно увеличивалась по сравнению с родительским штаммом.
На основе этого результата подтвердили то, что мутация ilvA(T381A, F383A) представляет собой эффективную мутацию для увеличения продукции изолейцина.
Референсный пример 3: Подтверждение продукции лейцина при помощи leuA(Р247С, R558H, G561D)
Референсный пример 3-1. Получение штамма CJL-8100
В частности, вектор pDCM2-leuA(R558H, G561D), содержащий мутации гена leuA, раскрытый в KR 10-2018-0077008 А, трансформировали в Corynebacterium glutamicum АТСС 13032 путем электропорации, и штамм, в котором вектор был встроен в хромосому путем рекомбинации гомологичной последовательности, был отобран в среде, содержащей 25 мг/л канамицина. Отобранный первичный штамм вновь подвергали вторичному кроссоверу, и отбирали штамм, в который были введены мутации гена leuA. Введение мутации в окончательно трансформированный штамм подтверждали путем осуществления ПЦР с использованием праймеров с SEQ ID NO: 55 и SEQ ID NO: 56 в условиях денатурации при 94°С в течение 5 минут с последующими 30 циклами денатурации при 94°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд, и затем полимеризации при 72°С в течение 5 минут и анализа нуклеотидных последовательностей, таким образом, подтверждая введение мутаций R558H и G561D. Штамм АТСС13032 leuA (R558H, G561D), трансформированный вектором pDCM2-leuA(R558H, G561D), был назван «CJL-8100».
Последовательности праймеров, используемых в референсном примере 3 ниже, представлены в таблице 18.


Референсный пример 3-2. Получение встраиваемого вектора, содержащего мутацию leuA
Вектор получали для встраивания мутации Р247С в CJL-8100, представляющий собой штамм, продуцирующий L-лейцин, в котором две мутации (R558H, G561D) введены в LeuA.
ПЦР осуществляли с использованием пар праймеров с SEQ ID NO: 57 и 58 или SEQ ID NO: 59 и 60, основанных на хромосоме штамма CJL-8100. ПЦР осуществляли в условиях денатурации при 94°С в течение 5 минут с последующими 30 циклами денатурации при 94°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд, и затем полимеризации при 72°С в течение 5 минут. Получающиеся в результате продукты ПЦР клонировали путем слияния между фрагментами ДНК с использованием линейного вектора pDCM2, расщепленного ферментом рестрикции SmaI, и фермента для слияния с получением вектора pDCM2-leuA(P247C, R558H, G561D) содержащего мутацию leuA, кодирующего мутант LeuA, при которой аргинин, представляющий собой 558-ю аминокислоту, заменен на гистидин, и глицин, представляющий собой 561-ю аминокислоту, заменен на аспарагиновую кислоту, и для замены пролина (Pro), представляющего собой 247-ю аминокислоту, на цистеин (Cys).
Референсный пример 3-3. Введение варианта leuA (Р247С) в штамм CJL-8100 и его оценка
Штамм, продуцирующий L-лейцин, CJL-8100, трансформировали в вектор pDCM2-leuA(P247C, R558H, G561D), полученный в референсном примере 3-2, и штамм, в котором вектор был встроен в хромосому путем рекомбинации гомологичной последовательности, был отобран в среде, содержащей 25 мг/л канамицина. Отобранный первичный штамм вновь подвергали вторичному кроссоверу, и отбирали штамм, в который была введена мутация гена-мишени. Введение мутации гена leuA в окончательно трансформированный штамм подтверждали путем осуществления ПЦР с использованием праймеров с SEQ ID NO: 55 и SEQ ID NO: 61 в условиях денатурации при 94°С в течение 5 минут с последующими 30 циклами денатурации при 94°С в течение 30 секунд, отжига при 55°С в течение 30 секунд и полимеризации при 72°С в течение 1 минуты и 30 секунд, и затем полимеризации при 72°С в течение 5 минут, и анализа нуклеотидных последовательностей. В результате анализа подтвердили то, что мутация leuA, кодирующая варианты LeuA (Р247С, R558H, G561D), в которых G, представляющий собой 1673-й нуклеотид гена leuA на хромосоме штамма, заменен на A, GC, представляющие собой 1682-й и 1683-й нуклеотиды, заменяли на AT, СС, представляющие собой 739-й и 740-й нуклеотиды, заменяли на TG, аргинин, представляющий собой 558-ю аминокислоту белка LeuA, заменен на гистидин, глицин, представляющий собой 561-ю аминокислоту, заменен на аспарагиновую кислоту, и пролин (Pro), представляющий собой 247-ю аминокислоту, заменен на цистеин (Cys).
Полученный таким образом CJL8100_leuA_P247C был назван «СА13-8105» и депонирован в Корейском центре культур микроорганизмов (KCCM), представляющем собой Международный депонирующий орган, в соответствии с Будапештским договором о международном признании депонирования микроорганизмов для целей патентной процедуры 29 апреля 2020 года под № KССМ12709Р.
Аминокислотная последовательность варианта LeuA (Р247С, R558H, G561D), включающего вышеприведенные три мутации, и нуклеотидная последовательность кодирующего его варианта leuA представлены в SEQ ID NO: 62 и SEQ ID NO: 63, соответственно.
Оценивали способность АТСС 13032 и полученных таким образом штаммов CJL-8100 и СА13-8105 продуцировать L-лейцин. В частности, культивирование в колбе осуществляли тем же самым образом, как в примере 2-1, и после завершения культивирования продукцию L-лейцина родительским штаммом и мутантными штаммами измеряли с использованием HPLC, и результаты представлены в таблице 19.

В соответствии с представленным в таблице 19 Corynebacterium glutamicum CJL8100, представляющий собой штамм, продуцирующий L-лейцин, продемонстрировал улучшенную способность продуцировать L-лейцин, приблизительно, на 130% по сравнению с родительским штаммом АТСС13032. Кроме того, штамм СА13-8105, в котором мутация LeuA_P247C дополнительно введена в штамм CJL8100, продемонстрировал улучшенную способность продуцировать L-лейцин, приблизительно, на 150% по сравнению с родительским штаммом CJL8100.
На основе этого результата подтвердили то, что мутация LeuA(R558H, G561D, Р247С) представляет собой эффективную мутацию для увеличения продукции лейцина.
Специалистам в данной области техники понятно, что настоящая заявка на изобретение может быть реализована в других конкретных формах без изменения его технической сущности или существенных характеристик. Описанные воплощения следует рассматривать во всех аспектах только как иллюстративные, а не ограничивающие. Таким образом, объем настоящей заявки на изобретение определен приложенной формулой изобретения, а не предшествующим описанием изобретения. Все изменения, которые находятся в пределах значения и диапазона эквивалентов формулы изобретения, охвачены объемом настоящей заявки на изобретение.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> CJ CheilJedang Corporation
<120> ВАРИАНТ АЛЬДОЛАЗЫ AROG И СПОСОБ ПРОДУКЦИИ АМИНОКИСЛОТЫ С
РАЗВЕТВЛЕННОЙ ЦЕПЬЮ С ЕГО ИСПОЛЬЗОВАНИЕМ
<130> OPA21303
<150> KR10-2021-0010911
<151> 2021-01-26
<160> 63
<170> KoPatentIn 3.0
<210> 1
<211> 466
<212> PRT
<213> Unknown
<220>
<223> aroG_WT
<400> 1
Met Asn Arg Gly Val Ser Trp Thr Val Asp Ile Pro Lys Glu Val Leu
1 5 10 15
Pro Asp Leu Pro Pro Leu Pro Glu Gly Met Gln Gln Gln Phe Glu Asp
20 25 30
Thr Ile Ser Arg Asp Ala Lys Gln Gln Pro Thr Trp Asp Arg Ala Gln
35 40 45
Ala Glu Asn Val Arg Lys Ile Leu Glu Ser Val Pro Pro Ile Val Val
50 55 60
Ala Pro Glu Val Leu Glu Leu Lys Gln Lys Leu Ala Asp Val Ala Asn
65 70 75 80
Gly Lys Ala Phe Leu Leu Gln Gly Gly Asp Cys Ala Glu Thr Phe Glu
85 90 95
Ser Asn Thr Glu Pro His Ile Arg Ala Asn Val Lys Thr Leu Leu Gln
100 105 110
Met Ala Val Val Leu Thr Tyr Gly Ala Ser Thr Pro Val Ile Lys Met
115 120 125
Ala Arg Ile Ala Gly Gln Tyr Ala Lys Pro Arg Ser Ser Asp Leu Asp
130 135 140
Gly Asn Gly Leu Pro Asn Tyr Arg Gly Asp Ile Val Asn Gly Val Glu
145 150 155 160
Ala Thr Pro Glu Ala Arg Arg His Asp Pro Ala Arg Met Ile Arg Ala
165 170 175
Tyr Ala Asn Ala Ser Ala Ala Met Asn Leu Val Arg Ala Leu Thr Ser
180 185 190
Ser Gly Thr Ala Asp Leu Tyr Arg Leu Ser Glu Trp Asn Arg Glu Phe
195 200 205
Val Ala Asn Ser Pro Ala Gly Ala Arg Tyr Glu Ala Leu Ala Arg Glu
210 215 220
Ile Asp Ser Gly Leu Arg Phe Met Glu Ala Cys Gly Val Ser Asp Glu
225 230 235 240
Ser Leu Arg Ala Ala Asp Ile Tyr Cys Ser His Glu Ala Leu Leu Val
245 250 255
Asp Tyr Glu Arg Ser Met Leu Arg Leu Ala Thr Asp Glu Glu Gly Asn
260 265 270
Glu Glu Leu Tyr Asp Leu Ser Ala His Gln Leu Trp Ile Gly Glu Arg
275 280 285
Thr Arg Gly Met Asp Asp Phe His Val Asn Phe Ala Ser Met Ile Ser
290 295 300
Asn Pro Ile Gly Ile Lys Ile Gly Pro Gly Ile Thr Pro Glu Glu Ala
305 310 315 320
Val Ala Tyr Ala Asp Lys Leu Asp Pro Asn Phe Glu Pro Gly Arg Leu
325 330 335
Thr Ile Val Ala Arg Met Gly His Asp Lys Val Arg Ser Val Leu Pro
340 345 350
Gly Val Ile Gln Ala Val Glu Ala Ser Gly His Lys Val Ile Trp Gln
355 360 365
Ser Asp Pro Met His Gly Asn Thr Phe Thr Ala Ser Asn Gly Tyr Lys
370 375 380
Thr Arg His Phe Asp Lys Val Ile Asp Glu Val Gln Gly Phe Phe Glu
385 390 395 400
Val His Arg Ala Leu Gly Thr His Pro Gly Gly Ile His Ile Glu Phe
405 410 415
Thr Gly Glu Asp Val Thr Glu Cys Leu Gly Gly Ala Glu Asp Ile Thr
420 425 430
Asp Val Asp Leu Pro Gly Arg Tyr Glu Ser Ala Cys Asp Pro Arg Leu
435 440 445
Asn Thr Gln Gln Ser Leu Glu Leu Ala Phe Leu Val Ala Glu Met Leu
450 455 460
Arg Asn
465
<210> 2
<211> 1401
<212> DNA
<213> Unknown
<220>
<223> aroG_WT
<400> 2
atgaataggg gtgtgagttg gacagttgat atccctaaag aagttctccc tgatttgcca 60
ccattgccag aaggcatgca gcagcagttc gaggacacca tttcccgtga cgctaagcag 120
caacctacgt gggatcgtgc acaggcagaa aacgtgcgca agatccttga gtcggttcct 180
ccaatcgttg ttgcccctga ggtacttgag ctgaagcaga agcttgctga tgttgccaac 240
ggtaaggcct tcctcttgca gggtggtgac tgtgcggaaa ctttcgagtc aaacactgag 300
ccgcacattc gcgccaacgt aaagactctg ctgcagatgg cagttgtttt gacctacggt 360
gcatccactc ctgtgatcaa gatggctcgt attgctggtc agtacgcaaa gcctcgctct 420
tctgatctgg atggaaatgg tctgccaaac taccgtggcg atatcgtcaa cggtgtggag 480
gcaaccccag aggctcgtcg ccacgatcct gcccgcatga tccgtgctta cgctaacgct 540
tctgctgcga tgaacttggt gcgcgcgctc accagctctg gcaccgctga tctttaccgt 600
ctcagcgagt ggaaccgcga gttcgttgcg aactccccag ctggtgcacg ctacgaggct 660
cttgctcgtg agatcgactc cggtctgcgc ttcatggaag catgtggcgt gtccgatgag 720
tccctgcgtg ctgcagatat ctactgctcc cacgaggctt tgctggtgga ttacgagcgt 780
tccatgctgc gtcttgcaac cgatgaggaa ggcaacgagg aactttacga tctttcagct 840
caccagctgt ggatcggcga gcgcacccgt ggcatggatg atttccatgt gaacttcgca 900
tccatgatct ctaacccaat cggcatcaag attggtcctg gtatcacccc tgaagaggct 960
gttgcatacg ctgacaagct cgatccgaac ttcgagcctg gccgtttgac catcgttgct 1020
cgcatgggcc acgacaaggt tcgctccgta cttcctggtg ttatccaggc tgttgaggca 1080
tccggacaca aggttatttg gcagtccgat ccgatgcacg gcaacacttt caccgcatcc 1140
aatggctaca agacccgtca cttcgacaag gttatcgatg aggtccaggg cttcttcgag 1200
gtccaccgcg cattgggcac ccacccaggc ggaatccaca ttgagttcac tggtgaagat 1260
gtcaccgagt gcctcggtgg cgctgaagac atcaccgatg ttgatctgcc aggccgctac 1320
gagtccgcat gcgatcctcg cctgaacact cagcagtctt tggagttggc tttcctcgtt 1380
gcagaaatgc tgcgtaacta a 1401
<210> 3
<211> 466
<212> PRT
<213> Artificial Sequence
<220>
<223> aroG_R217A
<400> 3
Met Asn Arg Gly Val Ser Trp Thr Val Asp Ile Pro Lys Glu Val Leu
1 5 10 15
Pro Asp Leu Pro Pro Leu Pro Glu Gly Met Gln Gln Gln Phe Glu Asp
20 25 30
Thr Ile Ser Arg Asp Ala Lys Gln Gln Pro Thr Trp Asp Arg Ala Gln
35 40 45
Ala Glu Asn Val Arg Lys Ile Leu Glu Ser Val Pro Pro Ile Val Val
50 55 60
Ala Pro Glu Val Leu Glu Leu Lys Gln Lys Leu Ala Asp Val Ala Asn
65 70 75 80
Gly Lys Ala Phe Leu Leu Gln Gly Gly Asp Cys Ala Glu Thr Phe Glu
85 90 95
Ser Asn Thr Glu Pro His Ile Arg Ala Asn Val Lys Thr Leu Leu Gln
100 105 110
Met Ala Val Val Leu Thr Tyr Gly Ala Ser Thr Pro Val Ile Lys Met
115 120 125
Ala Arg Ile Ala Gly Gln Tyr Ala Lys Pro Arg Ser Ser Asp Leu Asp
130 135 140
Gly Asn Gly Leu Pro Asn Tyr Arg Gly Asp Ile Val Asn Gly Val Glu
145 150 155 160
Ala Thr Pro Glu Ala Arg Arg His Asp Pro Ala Arg Met Ile Arg Ala
165 170 175
Tyr Ala Asn Ala Ser Ala Ala Met Asn Leu Val Arg Ala Leu Thr Ser
180 185 190
Ser Gly Thr Ala Asp Leu Tyr Arg Leu Ser Glu Trp Asn Arg Glu Phe
195 200 205
Val Ala Asn Ser Pro Ala Gly Ala Ala Tyr Glu Ala Leu Ala Arg Glu
210 215 220
Ile Asp Ser Gly Leu Arg Phe Met Glu Ala Cys Gly Val Ser Asp Glu
225 230 235 240
Ser Leu Arg Ala Ala Asp Ile Tyr Cys Ser His Glu Ala Leu Leu Val
245 250 255
Asp Tyr Glu Arg Ser Met Leu Arg Leu Ala Thr Asp Glu Glu Gly Asn
260 265 270
Glu Glu Leu Tyr Asp Leu Ser Ala His Gln Leu Trp Ile Gly Glu Arg
275 280 285
Thr Arg Gly Met Asp Asp Phe His Val Asn Phe Ala Ser Met Ile Ser
290 295 300
Asn Pro Ile Gly Ile Lys Ile Gly Pro Gly Ile Thr Pro Glu Glu Ala
305 310 315 320
Val Ala Tyr Ala Asp Lys Leu Asp Pro Asn Phe Glu Pro Gly Arg Leu
325 330 335
Thr Ile Val Ala Arg Met Gly His Asp Lys Val Arg Ser Val Leu Pro
340 345 350
Gly Val Ile Gln Ala Val Glu Ala Ser Gly His Lys Val Ile Trp Gln
355 360 365
Ser Asp Pro Met His Gly Asn Thr Phe Thr Ala Ser Asn Gly Tyr Lys
370 375 380
Thr Arg His Phe Asp Lys Val Ile Asp Glu Val Gln Gly Phe Phe Glu
385 390 395 400
Val His Arg Ala Leu Gly Thr His Pro Gly Gly Ile His Ile Glu Phe
405 410 415
Thr Gly Glu Asp Val Thr Glu Cys Leu Gly Gly Ala Glu Asp Ile Thr
420 425 430
Asp Val Asp Leu Pro Gly Arg Tyr Glu Ser Ala Cys Asp Pro Arg Leu
435 440 445
Asn Thr Gln Gln Ser Leu Glu Leu Ala Phe Leu Val Ala Glu Met Leu
450 455 460
Arg Asn
465
<210> 4
<211> 1401
<212> DNA
<213> Artificial Sequence
<220>
<223> aroG_R217A_nt
<400> 4
atgaataggg gtgtgagttg gacagttgat atccctaaag aagttctccc tgatttgcca 60
ccattgccag aaggcatgca gcagcagttc gaggacacca tttcccgtga cgctaagcag 120
caacctacgt gggatcgtgc acaggcagaa aacgtgcgca agatccttga gtcggttcct 180
ccaatcgttg ttgcccctga ggtacttgag ctgaagcaga agcttgctga tgttgccaac 240
ggtaaggcct tcctcttgca gggtggtgac tgtgcggaaa ctttcgagtc aaacactgag 300
ccgcacattc gcgccaacgt aaagactctg ctgcagatgg cagttgtttt gacctacggt 360
gcatccactc ctgtgatcaa gatggctcgt attgctggtc agtacgcaaa gcctcgctct 420
tctgatctgg atggaaatgg tctgccaaac taccgtggcg atatcgtcaa cggtgtggag 480
gcaaccccag aggctcgtcg ccacgatcct gcccgcatga tccgtgctta cgctaacgct 540
tctgctgcga tgaacttggt gcgcgcgctc accagctctg gcaccgctga tctttaccgt 600
ctcagcgagt ggaaccgcga gttcgttgcg aactccccag ctggtgcagc gtacgaggct 660
cttgctcgtg agatcgactc cggtctgcgc ttcatggaag catgtggcgt gtccgatgag 720
tccctgcgtg ctgcagatat ctactgctcc cacgaggctt tgctggtgga ttacgagcgt 780
tccatgctgc gtcttgcaac cgatgaggaa ggcaacgagg aactttacga tctttcagct 840
caccagctgt ggatcggcga gcgcacccgt ggcatggatg atttccatgt gaacttcgca 900
tccatgatct ctaacccaat cggcatcaag attggtcctg gtatcacccc tgaagaggct 960
gttgcatacg ctgacaagct cgatccgaac ttcgagcctg gccgtttgac catcgttgct 1020
cgcatgggcc acgacaaggt tcgctccgta cttcctggtg ttatccaggc tgttgaggca 1080
tccggacaca aggttatttg gcagtccgat ccgatgcacg gcaacacttt caccgcatcc 1140
aatggctaca agacccgtca cttcgacaag gttatcgatg aggtccaggg cttcttcgag 1200
gtccaccgcg cattgggcac ccacccaggc ggaatccaca ttgagttcac tggtgaagat 1260
gtcaccgagt gcctcggtgg cgctgaagac atcaccgatg ttgatctgcc aggccgctac 1320
gagtccgcat gcgatcctcg cctgaacact cagcagtctt tggagttggc tttcctcgtt 1380
gcagaaatgc tgcgtaacta a 1401
<210> 5
<211> 466
<212> PRT
<213> Artificial Sequence
<220>
<223> aroG_K310A
<400> 5
Met Asn Arg Gly Val Ser Trp Thr Val Asp Ile Pro Lys Glu Val Leu
1 5 10 15
Pro Asp Leu Pro Pro Leu Pro Glu Gly Met Gln Gln Gln Phe Glu Asp
20 25 30
Thr Ile Ser Arg Asp Ala Lys Gln Gln Pro Thr Trp Asp Arg Ala Gln
35 40 45
Ala Glu Asn Val Arg Lys Ile Leu Glu Ser Val Pro Pro Ile Val Val
50 55 60
Ala Pro Glu Val Leu Glu Leu Lys Gln Lys Leu Ala Asp Val Ala Asn
65 70 75 80
Gly Lys Ala Phe Leu Leu Gln Gly Gly Asp Cys Ala Glu Thr Phe Glu
85 90 95
Ser Asn Thr Glu Pro His Ile Arg Ala Asn Val Lys Thr Leu Leu Gln
100 105 110
Met Ala Val Val Leu Thr Tyr Gly Ala Ser Thr Pro Val Ile Lys Met
115 120 125
Ala Arg Ile Ala Gly Gln Tyr Ala Lys Pro Arg Ser Ser Asp Leu Asp
130 135 140
Gly Asn Gly Leu Pro Asn Tyr Arg Gly Asp Ile Val Asn Gly Val Glu
145 150 155 160
Ala Thr Pro Glu Ala Arg Arg His Asp Pro Ala Arg Met Ile Arg Ala
165 170 175
Tyr Ala Asn Ala Ser Ala Ala Met Asn Leu Val Arg Ala Leu Thr Ser
180 185 190
Ser Gly Thr Ala Asp Leu Tyr Arg Leu Ser Glu Trp Asn Arg Glu Phe
195 200 205
Val Ala Asn Ser Pro Ala Gly Ala Arg Tyr Glu Ala Leu Ala Arg Glu
210 215 220
Ile Asp Ser Gly Leu Arg Phe Met Glu Ala Cys Gly Val Ser Asp Glu
225 230 235 240
Ser Leu Arg Ala Ala Asp Ile Tyr Cys Ser His Glu Ala Leu Leu Val
245 250 255
Asp Tyr Glu Arg Ser Met Leu Arg Leu Ala Thr Asp Glu Glu Gly Asn
260 265 270
Glu Glu Leu Tyr Asp Leu Ser Ala His Gln Leu Trp Ile Gly Glu Arg
275 280 285
Thr Arg Gly Met Asp Asp Phe His Val Asn Phe Ala Ser Met Ile Ser
290 295 300
Asn Pro Ile Gly Ile Ala Ile Gly Pro Gly Ile Thr Pro Glu Glu Ala
305 310 315 320
Val Ala Tyr Ala Asp Lys Leu Asp Pro Asn Phe Glu Pro Gly Arg Leu
325 330 335
Thr Ile Val Ala Arg Met Gly His Asp Lys Val Arg Ser Val Leu Pro
340 345 350
Gly Val Ile Gln Ala Val Glu Ala Ser Gly His Lys Val Ile Trp Gln
355 360 365
Ser Asp Pro Met His Gly Asn Thr Phe Thr Ala Ser Asn Gly Tyr Lys
370 375 380
Thr Arg His Phe Asp Lys Val Ile Asp Glu Val Gln Gly Phe Phe Glu
385 390 395 400
Val His Arg Ala Leu Gly Thr His Pro Gly Gly Ile His Ile Glu Phe
405 410 415
Thr Gly Glu Asp Val Thr Glu Cys Leu Gly Gly Ala Glu Asp Ile Thr
420 425 430
Asp Val Asp Leu Pro Gly Arg Tyr Glu Ser Ala Cys Asp Pro Arg Leu
435 440 445
Asn Thr Gln Gln Ser Leu Glu Leu Ala Phe Leu Val Ala Glu Met Leu
450 455 460
Arg Asn
465
<210> 6
<211> 1401
<212> DNA
<213> Artificial Sequence
<220>
<223> aroG_K310A_nt
<400> 6
atgaataggg gtgtgagttg gacagttgat atccctaaag aagttctccc tgatttgcca 60
ccattgccag aaggcatgca gcagcagttc gaggacacca tttcccgtga cgctaagcag 120
caacctacgt gggatcgtgc acaggcagaa aacgtgcgca agatccttga gtcggttcct 180
ccaatcgttg ttgcccctga ggtacttgag ctgaagcaga agcttgctga tgttgccaac 240
ggtaaggcct tcctcttgca gggtggtgac tgtgcggaaa ctttcgagtc aaacactgag 300
ccgcacattc gcgccaacgt aaagactctg ctgcagatgg cagttgtttt gacctacggt 360
gcatccactc ctgtgatcaa gatggctcgt attgctggtc agtacgcaaa gcctcgctct 420
tctgatctgg atggaaatgg tctgccaaac taccgtggcg atatcgtcaa cggtgtggag 480
gcaaccccag aggctcgtcg ccacgatcct gcccgcatga tccgtgctta cgctaacgct 540
tctgctgcga tgaacttggt gcgcgcgctc accagctctg gcaccgctga tctttaccgt 600
ctcagcgagt ggaaccgcga gttcgttgcg aactccccag ctggtgcacg ctacgaggct 660
cttgctcgtg agatcgactc cggtctgcgc ttcatggaag catgtggcgt gtccgatgag 720
tccctgcgtg ctgcagatat ctactgctcc cacgaggctt tgctggtgga ttacgagcgt 780
tccatgctgc gtcttgcaac cgatgaggaa ggcaacgagg aactttacga tctttcagct 840
caccagctgt ggatcggcga gcgcacccgt ggcatggatg atttccatgt gaacttcgca 900
tccatgatct ctaacccaat cggcatcgcg attggtcctg gtatcacccc tgaagaggct 960
gttgcatacg ctgacaagct cgatccgaac ttcgagcctg gccgtttgac catcgttgct 1020
cgcatgggcc acgacaaggt tcgctccgta cttcctggtg ttatccaggc tgttgaggca 1080
tccggacaca aggttatttg gcagtccgat ccgatgcacg gcaacacttt caccgcatcc 1140
aatggctaca agacccgtca cttcgacaag gttatcgatg aggtccaggg cttcttcgag 1200
gtccaccgcg cattgggcac ccacccaggc ggaatccaca ttgagttcac tggtgaagat 1260
gtcaccgagt gcctcggtgg cgctgaagac atcaccgatg ttgatctgcc aggccgctac 1320
gagtccgcat gcgatcctcg cctgaacact cagcagtctt tggagttggc tttcctcgtt 1380
gcagaaatgc tgcgtaacta a 1401
<210> 7
<211> 466
<212> PRT
<213> Artificial Sequence
<220>
<223> aroG_R403A
<400> 7
Met Asn Arg Gly Val Ser Trp Thr Val Asp Ile Pro Lys Glu Val Leu
1 5 10 15
Pro Asp Leu Pro Pro Leu Pro Glu Gly Met Gln Gln Gln Phe Glu Asp
20 25 30
Thr Ile Ser Arg Asp Ala Lys Gln Gln Pro Thr Trp Asp Arg Ala Gln
35 40 45
Ala Glu Asn Val Arg Lys Ile Leu Glu Ser Val Pro Pro Ile Val Val
50 55 60
Ala Pro Glu Val Leu Glu Leu Lys Gln Lys Leu Ala Asp Val Ala Asn
65 70 75 80
Gly Lys Ala Phe Leu Leu Gln Gly Gly Asp Cys Ala Glu Thr Phe Glu
85 90 95
Ser Asn Thr Glu Pro His Ile Arg Ala Asn Val Lys Thr Leu Leu Gln
100 105 110
Met Ala Val Val Leu Thr Tyr Gly Ala Ser Thr Pro Val Ile Lys Met
115 120 125
Ala Arg Ile Ala Gly Gln Tyr Ala Lys Pro Arg Ser Ser Asp Leu Asp
130 135 140
Gly Asn Gly Leu Pro Asn Tyr Arg Gly Asp Ile Val Asn Gly Val Glu
145 150 155 160
Ala Thr Pro Glu Ala Arg Arg His Asp Pro Ala Arg Met Ile Arg Ala
165 170 175
Tyr Ala Asn Ala Ser Ala Ala Met Asn Leu Val Arg Ala Leu Thr Ser
180 185 190
Ser Gly Thr Ala Asp Leu Tyr Arg Leu Ser Glu Trp Asn Arg Glu Phe
195 200 205
Val Ala Asn Ser Pro Ala Gly Ala Arg Tyr Glu Ala Leu Ala Arg Glu
210 215 220
Ile Asp Ser Gly Leu Arg Phe Met Glu Ala Cys Gly Val Ser Asp Glu
225 230 235 240
Ser Leu Arg Ala Ala Asp Ile Tyr Cys Ser His Glu Ala Leu Leu Val
245 250 255
Asp Tyr Glu Arg Ser Met Leu Arg Leu Ala Thr Asp Glu Glu Gly Asn
260 265 270
Glu Glu Leu Tyr Asp Leu Ser Ala His Gln Leu Trp Ile Gly Glu Arg
275 280 285
Thr Arg Gly Met Asp Asp Phe His Val Asn Phe Ala Ser Met Ile Ser
290 295 300
Asn Pro Ile Gly Ile Lys Ile Gly Pro Gly Ile Thr Pro Glu Glu Ala
305 310 315 320
Val Ala Tyr Ala Asp Lys Leu Asp Pro Asn Phe Glu Pro Gly Arg Leu
325 330 335
Thr Ile Val Ala Arg Met Gly His Asp Lys Val Arg Ser Val Leu Pro
340 345 350
Gly Val Ile Gln Ala Val Glu Ala Ser Gly His Lys Val Ile Trp Gln
355 360 365
Ser Asp Pro Met His Gly Asn Thr Phe Thr Ala Ser Asn Gly Tyr Lys
370 375 380
Thr Arg His Phe Asp Lys Val Ile Asp Glu Val Gln Gly Phe Phe Glu
385 390 395 400
Val His Ala Ala Leu Gly Thr His Pro Gly Gly Ile His Ile Glu Phe
405 410 415
Thr Gly Glu Asp Val Thr Glu Cys Leu Gly Gly Ala Glu Asp Ile Thr
420 425 430
Asp Val Asp Leu Pro Gly Arg Tyr Glu Ser Ala Cys Asp Pro Arg Leu
435 440 445
Asn Thr Gln Gln Ser Leu Glu Leu Ala Phe Leu Val Ala Glu Met Leu
450 455 460
Arg Asn
465
<210> 8
<211> 1401
<212> DNA
<213> Artificial Sequence
<220>
<223> aroG_R403A_nt
<400> 8
atgaataggg gtgtgagttg gacagttgat atccctaaag aagttctccc tgatttgcca 60
ccattgccag aaggcatgca gcagcagttc gaggacacca tttcccgtga cgctaagcag 120
caacctacgt gggatcgtgc acaggcagaa aacgtgcgca agatccttga gtcggttcct 180
ccaatcgttg ttgcccctga ggtacttgag ctgaagcaga agcttgctga tgttgccaac 240
ggtaaggcct tcctcttgca gggtggtgac tgtgcggaaa ctttcgagtc aaacactgag 300
ccgcacattc gcgccaacgt aaagactctg ctgcagatgg cagttgtttt gacctacggt 360
gcatccactc ctgtgatcaa gatggctcgt attgctggtc agtacgcaaa gcctcgctct 420
tctgatctgg atggaaatgg tctgccaaac taccgtggcg atatcgtcaa cggtgtggag 480
gcaaccccag aggctcgtcg ccacgatcct gcccgcatga tccgtgctta cgctaacgct 540
tctgctgcga tgaacttggt gcgcgcgctc accagctctg gcaccgctga tctttaccgt 600
ctcagcgagt ggaaccgcga gttcgttgcg aactccccag ctggtgcacg ctacgaggct 660
cttgctcgtg agatcgactc cggtctgcgc ttcatggaag catgtggcgt gtccgatgag 720
tccctgcgtg ctgcagatat ctactgctcc cacgaggctt tgctggtgga ttacgagcgt 780
tccatgctgc gtcttgcaac cgatgaggaa ggcaacgagg aactttacga tctttcagct 840
caccagctgt ggatcggcga gcgcacccgt ggcatggatg atttccatgt gaacttcgca 900
tccatgatct ctaacccaat cggcatcaag attggtcctg gtatcacccc tgaagaggct 960
gttgcatacg ctgacaagct cgatccgaac ttcgagcctg gccgtttgac catcgttgct 1020
cgcatgggcc acgacaaggt tcgctccgta cttcctggtg ttatccaggc tgttgaggca 1080
tccggacaca aggttatttg gcagtccgat ccgatgcacg gcaacacttt caccgcatcc 1140
aatggctaca agacccgtca cttcgacaag gttatcgatg aggtccaggg cttcttcgag 1200
gtccacgcgg cattgggcac ccacccaggc ggaatccaca ttgagttcac tggtgaagat 1260
gtcaccgagt gcctcggtgg cgctgaagac atcaccgatg ttgatctgcc aggccgctac 1320
gagtccgcat gcgatcctcg cctgaacact cagcagtctt tggagttggc tttcctcgtt 1380
gcagaaatgc tgcgtaacta a 1401
<210> 9
<211> 466
<212> PRT
<213> Artificial Sequence
<220>
<223> aroG_E462A
<400> 9
Met Asn Arg Gly Val Ser Trp Thr Val Asp Ile Pro Lys Glu Val Leu
1 5 10 15
Pro Asp Leu Pro Pro Leu Pro Glu Gly Met Gln Gln Gln Phe Glu Asp
20 25 30
Thr Ile Ser Arg Asp Ala Lys Gln Gln Pro Thr Trp Asp Arg Ala Gln
35 40 45
Ala Glu Asn Val Arg Lys Ile Leu Glu Ser Val Pro Pro Ile Val Val
50 55 60
Ala Pro Glu Val Leu Glu Leu Lys Gln Lys Leu Ala Asp Val Ala Asn
65 70 75 80
Gly Lys Ala Phe Leu Leu Gln Gly Gly Asp Cys Ala Glu Thr Phe Glu
85 90 95
Ser Asn Thr Glu Pro His Ile Arg Ala Asn Val Lys Thr Leu Leu Gln
100 105 110
Met Ala Val Val Leu Thr Tyr Gly Ala Ser Thr Pro Val Ile Lys Met
115 120 125
Ala Arg Ile Ala Gly Gln Tyr Ala Lys Pro Arg Ser Ser Asp Leu Asp
130 135 140
Gly Asn Gly Leu Pro Asn Tyr Arg Gly Asp Ile Val Asn Gly Val Glu
145 150 155 160
Ala Thr Pro Glu Ala Arg Arg His Asp Pro Ala Arg Met Ile Arg Ala
165 170 175
Tyr Ala Asn Ala Ser Ala Ala Met Asn Leu Val Arg Ala Leu Thr Ser
180 185 190
Ser Gly Thr Ala Asp Leu Tyr Arg Leu Ser Glu Trp Asn Arg Glu Phe
195 200 205
Val Ala Asn Ser Pro Ala Gly Ala Arg Tyr Glu Ala Leu Ala Arg Glu
210 215 220
Ile Asp Ser Gly Leu Arg Phe Met Glu Ala Cys Gly Val Ser Asp Glu
225 230 235 240
Ser Leu Arg Ala Ala Asp Ile Tyr Cys Ser His Glu Ala Leu Leu Val
245 250 255
Asp Tyr Glu Arg Ser Met Leu Arg Leu Ala Thr Asp Glu Glu Gly Asn
260 265 270
Glu Glu Leu Tyr Asp Leu Ser Ala His Gln Leu Trp Ile Gly Glu Arg
275 280 285
Thr Arg Gly Met Asp Asp Phe His Val Asn Phe Ala Ser Met Ile Ser
290 295 300
Asn Pro Ile Gly Ile Lys Ile Gly Pro Gly Ile Thr Pro Glu Glu Ala
305 310 315 320
Val Ala Tyr Ala Asp Lys Leu Asp Pro Asn Phe Glu Pro Gly Arg Leu
325 330 335
Thr Ile Val Ala Arg Met Gly His Asp Lys Val Arg Ser Val Leu Pro
340 345 350
Gly Val Ile Gln Ala Val Glu Ala Ser Gly His Lys Val Ile Trp Gln
355 360 365
Ser Asp Pro Met His Gly Asn Thr Phe Thr Ala Ser Asn Gly Tyr Lys
370 375 380
Thr Arg His Phe Asp Lys Val Ile Asp Glu Val Gln Gly Phe Phe Glu
385 390 395 400
Val His Arg Ala Leu Gly Thr His Pro Gly Gly Ile His Ile Glu Phe
405 410 415
Thr Gly Glu Asp Val Thr Glu Cys Leu Gly Gly Ala Glu Asp Ile Thr
420 425 430
Asp Val Asp Leu Pro Gly Arg Tyr Glu Ser Ala Cys Asp Pro Arg Leu
435 440 445
Asn Thr Gln Gln Ser Leu Glu Leu Ala Phe Leu Val Ala Ala Met Leu
450 455 460
Arg Asn
465
<210> 10
<211> 1401
<212> DNA
<213> Artificial Sequence
<220>
<223> aroG_E462A_nt
<400> 10
atgaataggg gtgtgagttg gacagttgat atccctaaag aagttctccc tgatttgcca 60
ccattgccag aaggcatgca gcagcagttc gaggacacca tttcccgtga cgctaagcag 120
caacctacgt gggatcgtgc acaggcagaa aacgtgcgca agatccttga gtcggttcct 180
ccaatcgttg ttgcccctga ggtacttgag ctgaagcaga agcttgctga tgttgccaac 240
ggtaaggcct tcctcttgca gggtggtgac tgtgcggaaa ctttcgagtc aaacactgag 300
ccgcacattc gcgccaacgt aaagactctg ctgcagatgg cagttgtttt gacctacggt 360
gcatccactc ctgtgatcaa gatggctcgt attgctggtc agtacgcaaa gcctcgctct 420
tctgatctgg atggaaatgg tctgccaaac taccgtggcg atatcgtcaa cggtgtggag 480
gcaaccccag aggctcgtcg ccacgatcct gcccgcatga tccgtgctta cgctaacgct 540
tctgctgcga tgaacttggt gcgcgcgctc accagctctg gcaccgctga tctttaccgt 600
ctcagcgagt ggaaccgcga gttcgttgcg aactccccag ctggtgcacg ctacgaggct 660
cttgctcgtg agatcgactc cggtctgcgc ttcatggaag catgtggcgt gtccgatgag 720
tccctgcgtg ctgcagatat ctactgctcc cacgaggctt tgctggtgga ttacgagcgt 780
tccatgctgc gtcttgcaac cgatgaggaa ggcaacgagg aactttacga tctttcagct 840
caccagctgt ggatcggcga gcgcacccgt ggcatggatg atttccatgt gaacttcgca 900
tccatgatct ctaacccaat cggcatcaag attggtcctg gtatcacccc tgaagaggct 960
gttgcatacg ctgacaagct cgatccgaac ttcgagcctg gccgtttgac catcgttgct 1020
cgcatgggcc acgacaaggt tcgctccgta cttcctggtg ttatccaggc tgttgaggca 1080
tccggacaca aggttatttg gcagtccgat ccgatgcacg gcaacacttt caccgcatcc 1140
aatggctaca agacccgtca cttcgacaag gttatcgatg aggtccaggg cttcttcgag 1200
gtccaccgcg cattgggcac ccacccaggc ggaatccaca ttgagttcac tggtgaagat 1260
gtcaccgagt gcctcggtgg cgctgaagac atcaccgatg ttgatctgcc aggccgctac 1320
gagtccgcat gcgatcctcg cctgaacact cagcagtctt tggagttggc tttcctcgtt 1380
gcagcgatgc tgcgtaacta a 1401
<210> 11
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 11
tgatgcgcgt cataatttag 20
<210> 12
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 12
ctcatctctg actggacgtg 20
<210> 13
<211> 41
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 13
gtgaattcga gctcggtacc cagttgaatg ctaccaactt g 41
<210> 14
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 14
cacgagcaag agcctcgtac gctgcaccag ctggg 35
<210> 15
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 15
ggtcgactct agaggatccc cgctgttatt actgtgcctg 40
<210> 16
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 16
gaactcccca gctggtgcag cgtacgaggc tcttgctcg 39
<210> 17
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 17
gggtgatacc aggaccaatc gcgatgccga ttgggttag 39
<210> 18
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 18
ctctaaccca atcggcatcg cgattggtcc tggtatcac 39
<210> 19
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 19
ctgggtgggt gcccaatgcc gcgtggacct cgaagaag 38
<210> 20
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 20
gggcttcttc gaggtccacg cggcattggg cacccac 37
<210> 21
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 21
aagcttagtt acgcagcatc gctgcaacga ggaaagcc 38
<210> 22
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 22
gttggctttc ctcgttgcag cgatgctgcg taactaagc 39
<210> 23
<211> 466
<212> PRT
<213> Artificial Sequence
<220>
<223> aroG_R217A+K310A+E462A
<400> 23
Met Asn Arg Gly Val Ser Trp Thr Val Asp Ile Pro Lys Glu Val Leu
1 5 10 15
Pro Asp Leu Pro Pro Leu Pro Glu Gly Met Gln Gln Gln Phe Glu Asp
20 25 30
Thr Ile Ser Arg Asp Ala Lys Gln Gln Pro Thr Trp Asp Arg Ala Gln
35 40 45
Ala Glu Asn Val Arg Lys Ile Leu Glu Ser Val Pro Pro Ile Val Val
50 55 60
Ala Pro Glu Val Leu Glu Leu Lys Gln Lys Leu Ala Asp Val Ala Asn
65 70 75 80
Gly Lys Ala Phe Leu Leu Gln Gly Gly Asp Cys Ala Glu Thr Phe Glu
85 90 95
Ser Asn Thr Glu Pro His Ile Arg Ala Asn Val Lys Thr Leu Leu Gln
100 105 110
Met Ala Val Val Leu Thr Tyr Gly Ala Ser Thr Pro Val Ile Lys Met
115 120 125
Ala Arg Ile Ala Gly Gln Tyr Ala Lys Pro Arg Ser Ser Asp Leu Asp
130 135 140
Gly Asn Gly Leu Pro Asn Tyr Arg Gly Asp Ile Val Asn Gly Val Glu
145 150 155 160
Ala Thr Pro Glu Ala Arg Arg His Asp Pro Ala Arg Met Ile Arg Ala
165 170 175
Tyr Ala Asn Ala Ser Ala Ala Met Asn Leu Val Arg Ala Leu Thr Ser
180 185 190
Ser Gly Thr Ala Asp Leu Tyr Arg Leu Ser Glu Trp Asn Arg Glu Phe
195 200 205
Val Ala Asn Ser Pro Ala Gly Ala Ala Tyr Glu Ala Leu Ala Arg Glu
210 215 220
Ile Asp Ser Gly Leu Arg Phe Met Glu Ala Cys Gly Val Ser Asp Glu
225 230 235 240
Ser Leu Arg Ala Ala Asp Ile Tyr Cys Ser His Glu Ala Leu Leu Val
245 250 255
Asp Tyr Glu Arg Ser Met Leu Arg Leu Ala Thr Asp Glu Glu Gly Asn
260 265 270
Glu Glu Leu Tyr Asp Leu Ser Ala His Gln Leu Trp Ile Gly Glu Arg
275 280 285
Thr Arg Gly Met Asp Asp Phe His Val Asn Phe Ala Ser Met Ile Ser
290 295 300
Asn Pro Ile Gly Ile Ala Ile Gly Pro Gly Ile Thr Pro Glu Glu Ala
305 310 315 320
Val Ala Tyr Ala Asp Lys Leu Asp Pro Asn Phe Glu Pro Gly Arg Leu
325 330 335
Thr Ile Val Ala Arg Met Gly His Asp Lys Val Arg Ser Val Leu Pro
340 345 350
Gly Val Ile Gln Ala Val Glu Ala Ser Gly His Lys Val Ile Trp Gln
355 360 365
Ser Asp Pro Met His Gly Asn Thr Phe Thr Ala Ser Asn Gly Tyr Lys
370 375 380
Thr Arg His Phe Asp Lys Val Ile Asp Glu Val Gln Gly Phe Phe Glu
385 390 395 400
Val His Arg Ala Leu Gly Thr His Pro Gly Gly Ile His Ile Glu Phe
405 410 415
Thr Gly Glu Asp Val Thr Glu Cys Leu Gly Gly Ala Glu Asp Ile Thr
420 425 430
Asp Val Asp Leu Pro Gly Arg Tyr Glu Ser Ala Cys Asp Pro Arg Leu
435 440 445
Asn Thr Gln Gln Ser Leu Glu Leu Ala Phe Leu Val Ala Ala Met Leu
450 455 460
Arg Asn
465
<210> 24
<211> 1401
<212> DNA
<213> Artificial Sequence
<220>
<223> aroG_R217A+K310A+E462A_nt
<400> 24
atgaataggg gtgtgagttg gacagttgat atccctaaag aagttctccc tgatttgcca 60
ccattgccag aaggcatgca gcagcagttc gaggacacca tttcccgtga cgctaagcag 120
caacctacgt gggatcgtgc acaggcagaa aacgtgcgca agatccttga gtcggttcct 180
ccaatcgttg ttgcccctga ggtacttgag ctgaagcaga agcttgctga tgttgccaac 240
ggtaaggcct tcctcttgca gggtggtgac tgtgcggaaa ctttcgagtc aaacactgag 300
ccgcacattc gcgccaacgt aaagactctg ctgcagatgg cagttgtttt gacctacggt 360
gcatccactc ctgtgatcaa gatggctcgt attgctggtc agtacgcaaa gcctcgctct 420
tctgatctgg atggaaatgg tctgccaaac taccgtggcg atatcgtcaa cggtgtggag 480
gcaaccccag aggctcgtcg ccacgatcct gcccgcatga tccgtgctta cgctaacgct 540
tctgctgcga tgaacttggt gcgcgcgctc accagctctg gcaccgctga tctttaccgt 600
ctcagcgagt ggaaccgcga gttcgttgcg aactccccag ctggtgcagc gtacgaggct 660
cttgctcgtg agatcgactc cggtctgcgc ttcatggaag catgtggcgt gtccgatgag 720
tccctgcgtg ctgcagatat ctactgctcc cacgaggctt tgctggtgga ttacgagcgt 780
tccatgctgc gtcttgcaac cgatgaggaa ggcaacgagg aactttacga tctttcagct 840
caccagctgt ggatcggcga gcgcacccgt ggcatggatg atttccatgt gaacttcgca 900
tccatgatct ctaacccaat cggcatcgcg attggtcctg gtatcacccc tgaagaggct 960
gttgcatacg ctgacaagct cgatccgaac ttcgagcctg gccgtttgac catcgttgct 1020
cgcatgggcc acgacaaggt tcgctccgta cttcctggtg ttatccaggc tgttgaggca 1080
tccggacaca aggttatttg gcagtccgat ccgatgcacg gcaacacttt caccgcatcc 1140
aatggctaca agacccgtca cttcgacaag gttatcgatg aggtccaggg cttcttcgag 1200
gtccaccgcg cattgggcac ccacccaggc ggaatccaca ttgagttcac tggtgaagat 1260
gtcaccgagt gcctcggtgg cgctgaagac atcaccgatg ttgatctgcc aggccgctac 1320
gagtccgcat gcgatcctcg cctgaacact cagcagtctt tggagttggc tttcctcgtt 1380
gcagcgatgc tgcgtaacta a 1401
<210> 25
<211> 466
<212> PRT
<213> Artificial Sequence
<220>
<223> aroG_R217A+K310A+R403A+E462A
<400> 25
Met Asn Arg Gly Val Ser Trp Thr Val Asp Ile Pro Lys Glu Val Leu
1 5 10 15
Pro Asp Leu Pro Pro Leu Pro Glu Gly Met Gln Gln Gln Phe Glu Asp
20 25 30
Thr Ile Ser Arg Asp Ala Lys Gln Gln Pro Thr Trp Asp Arg Ala Gln
35 40 45
Ala Glu Asn Val Arg Lys Ile Leu Glu Ser Val Pro Pro Ile Val Val
50 55 60
Ala Pro Glu Val Leu Glu Leu Lys Gln Lys Leu Ala Asp Val Ala Asn
65 70 75 80
Gly Lys Ala Phe Leu Leu Gln Gly Gly Asp Cys Ala Glu Thr Phe Glu
85 90 95
Ser Asn Thr Glu Pro His Ile Arg Ala Asn Val Lys Thr Leu Leu Gln
100 105 110
Met Ala Val Val Leu Thr Tyr Gly Ala Ser Thr Pro Val Ile Lys Met
115 120 125
Ala Arg Ile Ala Gly Gln Tyr Ala Lys Pro Arg Ser Ser Asp Leu Asp
130 135 140
Gly Asn Gly Leu Pro Asn Tyr Arg Gly Asp Ile Val Asn Gly Val Glu
145 150 155 160
Ala Thr Pro Glu Ala Arg Arg His Asp Pro Ala Arg Met Ile Arg Ala
165 170 175
Tyr Ala Asn Ala Ser Ala Ala Met Asn Leu Val Arg Ala Leu Thr Ser
180 185 190
Ser Gly Thr Ala Asp Leu Tyr Arg Leu Ser Glu Trp Asn Arg Glu Phe
195 200 205
Val Ala Asn Ser Pro Ala Gly Ala Ala Tyr Glu Ala Leu Ala Arg Glu
210 215 220
Ile Asp Ser Gly Leu Arg Phe Met Glu Ala Cys Gly Val Ser Asp Glu
225 230 235 240
Ser Leu Arg Ala Ala Asp Ile Tyr Cys Ser His Glu Ala Leu Leu Val
245 250 255
Asp Tyr Glu Arg Ser Met Leu Arg Leu Ala Thr Asp Glu Glu Gly Asn
260 265 270
Glu Glu Leu Tyr Asp Leu Ser Ala His Gln Leu Trp Ile Gly Glu Arg
275 280 285
Thr Arg Gly Met Asp Asp Phe His Val Asn Phe Ala Ser Met Ile Ser
290 295 300
Asn Pro Ile Gly Ile Ala Ile Gly Pro Gly Ile Thr Pro Glu Glu Ala
305 310 315 320
Val Ala Tyr Ala Asp Lys Leu Asp Pro Asn Phe Glu Pro Gly Arg Leu
325 330 335
Thr Ile Val Ala Arg Met Gly His Asp Lys Val Arg Ser Val Leu Pro
340 345 350
Gly Val Ile Gln Ala Val Glu Ala Ser Gly His Lys Val Ile Trp Gln
355 360 365
Ser Asp Pro Met His Gly Asn Thr Phe Thr Ala Ser Asn Gly Tyr Lys
370 375 380
Thr Arg His Phe Asp Lys Val Ile Asp Glu Val Gln Gly Phe Phe Glu
385 390 395 400
Val His Ala Ala Leu Gly Thr His Pro Gly Gly Ile His Ile Glu Phe
405 410 415
Thr Gly Glu Asp Val Thr Glu Cys Leu Gly Gly Ala Glu Asp Ile Thr
420 425 430
Asp Val Asp Leu Pro Gly Arg Tyr Glu Ser Ala Cys Asp Pro Arg Leu
435 440 445
Asn Thr Gln Gln Ser Leu Glu Leu Ala Phe Leu Val Ala Ala Met Leu
450 455 460
Arg Asn
465
<210> 26
<211> 1401
<212> DNA
<213> Artificial Sequence
<220>
<223> aroG_R217A+K310A+R403A+E462A_nt
<400> 26
atgaataggg gtgtgagttg gacagttgat atccctaaag aagttctccc tgatttgcca 60
ccattgccag aaggcatgca gcagcagttc gaggacacca tttcccgtga cgctaagcag 120
caacctacgt gggatcgtgc acaggcagaa aacgtgcgca agatccttga gtcggttcct 180
ccaatcgttg ttgcccctga ggtacttgag ctgaagcaga agcttgctga tgttgccaac 240
ggtaaggcct tcctcttgca gggtggtgac tgtgcggaaa ctttcgagtc aaacactgag 300
ccgcacattc gcgccaacgt aaagactctg ctgcagatgg cagttgtttt gacctacggt 360
gcatccactc ctgtgatcaa gatggctcgt attgctggtc agtacgcaaa gcctcgctct 420
tctgatctgg atggaaatgg tctgccaaac taccgtggcg atatcgtcaa cggtgtggag 480
gcaaccccag aggctcgtcg ccacgatcct gcccgcatga tccgtgctta cgctaacgct 540
tctgctgcga tgaacttggt gcgcgcgctc accagctctg gcaccgctga tctttaccgt 600
ctcagcgagt ggaaccgcga gttcgttgcg aactccccag ctggtgcagc gtacgaggct 660
cttgctcgtg agatcgactc cggtctgcgc ttcatggaag catgtggcgt gtccgatgag 720
tccctgcgtg ctgcagatat ctactgctcc cacgaggctt tgctggtgga ttacgagcgt 780
tccatgctgc gtcttgcaac cgatgaggaa ggcaacgagg aactttacga tctttcagct 840
caccagctgt ggatcggcga gcgcacccgt ggcatggatg atttccatgt gaacttcgca 900
tccatgatct ctaacccaat cggcatcgcg attggtcctg gtatcacccc tgaagaggct 960
gttgcatacg ctgacaagct cgatccgaac ttcgagcctg gccgtttgac catcgttgct 1020
cgcatgggcc acgacaaggt tcgctccgta cttcctggtg ttatccaggc tgttgaggca 1080
tccggacaca aggttatttg gcagtccgat ccgatgcacg gcaacacttt caccgcatcc 1140
aatggctaca agacccgtca cttcgacaag gttatcgatg aggtccaggg cttcttcgag 1200
gtccacgcgg cattgggcac ccacccaggc ggaatccaca ttgagttcac tggtgaagat 1260
gtcaccgagt gcctcggtgg cgctgaagac atcaccgatg ttgatctgcc aggccgctac 1320
gagtccgcat gcgatcctcg cctgaacact cagcagtctt tggagttggc tttcctcgtt 1380
gcagcgatgc tgcgtaacta a 1401
<210> 27
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 27
tatgcttcac cacatgactt c 21
<210> 28
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 28
aaatcatttg agaaaactcg agg 23
<210> 29
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 29
gtcacccgat cgtctgaag 19
<210> 30
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 30
gtcttaaaac cggttgat 18
<210> 31
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 31
caatgctggc tgcgtacgc 19
<210> 32
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 32
ctcctcgcga ggaaccaact 20
<210> 33
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 33
tcgagctcgg taccccgctt ttgcactcat cgagc 35
<210> 34
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 34
cacgatcaga tgtgcatcat cat 23
<210> 35
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 35
atgatgatgc acatctgatc gtg 23
<210> 36
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 36
ctctagagga tccccgagca tcttccaaaa ccttg 35
<210> 37
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 37
tcgagctcgg tacccatgag tgaaacatac gtgtc 35
<210> 38
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 38
gcgcttgagg tactctgcca gcgcgatgtc atcatccgg 39
<210> 39
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 39
ccggatgatg acatcgcgct ggcagagtac ctcaagcgc 39
<210> 40
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 40
ctctagagga tcccccgtca ccgacacctc caca 34
<210> 41
<211> 437
<212> PRT
<213> Artificial Sequence
<220>
<223> gltA_M312I
<400> 41
Met Phe Glu Arg Asp Ile Val Ala Thr Asp Asn Asn Lys Ala Val Leu
1 5 10 15
His Tyr Pro Gly Gly Glu Phe Glu Met Asp Ile Ile Glu Ala Ser Glu
20 25 30
Gly Asn Asn Gly Val Val Leu Gly Lys Met Leu Ser Glu Thr Gly Leu
35 40 45
Ile Thr Phe Asp Pro Gly Tyr Val Ser Thr Gly Ser Thr Glu Ser Lys
50 55 60
Ile Thr Tyr Ile Asp Gly Asp Ala Gly Ile Leu Arg Tyr Arg Gly Tyr
65 70 75 80
Asp Ile Ala Asp Leu Ala Glu Asn Ala Thr Phe Asn Glu Val Ser Tyr
85 90 95
Leu Leu Ile Asn Gly Glu Leu Pro Thr Pro Asp Glu Leu His Lys Phe
100 105 110
Asn Asp Glu Ile Arg His His Thr Leu Leu Asp Glu Asp Phe Lys Ser
115 120 125
Gln Phe Asn Val Phe Pro Arg Asp Ala His Pro Met Ala Thr Leu Ala
130 135 140
Ser Ser Val Asn Ile Leu Ser Thr Tyr Tyr Gln Asp Gln Leu Asn Pro
145 150 155 160
Leu Asp Glu Ala Gln Leu Asp Lys Ala Thr Val Arg Leu Met Ala Lys
165 170 175
Val Pro Met Leu Ala Ala Tyr Ala His Arg Ala Arg Lys Gly Ala Pro
180 185 190
Tyr Met Tyr Pro Asp Asn Ser Leu Asn Ala Arg Glu Asn Phe Leu Arg
195 200 205
Met Met Phe Gly Tyr Pro Thr Glu Pro Tyr Glu Ile Asp Pro Ile Met
210 215 220
Val Lys Ala Leu Asp Lys Leu Leu Ile Leu His Ala Asp His Glu Gln
225 230 235 240
Asn Cys Ser Thr Ser Thr Val Arg Met Ile Gly Ser Ala Gln Ala Asn
245 250 255
Met Phe Val Ser Ile Ala Gly Gly Ile Asn Ala Leu Ser Gly Pro Leu
260 265 270
His Gly Gly Ala Asn Gln Ala Val Leu Glu Met Leu Glu Asp Ile Lys
275 280 285
Ser Asn His Gly Gly Asp Ala Thr Glu Phe Met Asn Lys Val Lys Asn
290 295 300
Lys Glu Asp Gly Val Arg Leu Ile Gly Phe Gly His Arg Val Tyr Lys
305 310 315 320
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
325 330 335
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
340 345 350
Glu Glu Ile Ala Leu Ala Asp Asp Tyr Phe Ile Ser Arg Lys Leu Tyr
355 360 365
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
370 375 380
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
385 390 395 400
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn Lys Ile
405 410 415
Asn Arg Pro Arg Gln Val Tyr Thr Gly Asn Glu Ser Arg Lys Leu Val
420 425 430
Pro Arg Glu Glu Arg
435
<210> 42
<211> 1314
<212> DNA
<213> Artificial Sequence
<220>
<223> gltA_M312I_nt
<400> 42
atgtttgaaa gggatatcgt ggctactgat aacaacaagg ctgtcctgca ctaccccggt 60
ggcgagttcg aaatggacat catcgaggct tctgagggta acaacggtgt tgtcctgggc 120
aagatgctgt ctgagactgg actgatcact tttgacccag gttatgtgag cactggctcc 180
accgagtcga agatcaccta catcgatggc gatgcgggaa tcctgcgtta ccgcggctat 240
gacatcgctg atctggctga gaatgccacc ttcaacgagg tttcttacct acttatcaac 300
ggtgagctac caaccccaga tgagcttcac aagtttaacg acgagattcg ccaccacacc 360
cttctggacg aggacttcaa gtcccagttc aacgtgttcc cacgcgacgc tcacccaatg 420
gcaaccttgg cttcctcggt taacattttg tctacctact accaggacca gctgaaccca 480
ctcgatgagg cacagcttga taaggcaacc gttcgcctca tggcaaaggt tccaatgctg 540
gctgcgtacg cacaccgcgc acgcaagggt gctccttaca tgtacccaga caactccctc 600
aatgcgcgtg agaacttcct gcgcatgatg ttcggttacc caaccgagcc atacgagatc 660
gacccaatca tggtcaaggc tctggacaag ctgctcatcc tgcacgctga ccacgagcag 720
aactgctcca cctccaccgt tcgtatgatc ggttccgcac aggccaacat gtttgtctcc 780
atcgctggtg gcatcaacgc tctgtccggc ccactgcacg gtggcgcaaa ccaggctgtt 840
ctggagatgc tcgaagacat caagagcaac cacggtggcg acgcaaccga gttcatgaac 900
aaggtcaaga acaaggaaga cggcgtccgc ctcatcggct tcggacaccg cgtttacaag 960
aactacgatc cacgtgcagc aatcgtcaag gagaccgcac acgagatcct cgagcacctc 1020
ggtggcgacg atcttctgga tctggcaatc aagctggaag aaattgcact ggctgatgat 1080
tacttcatct cccgcaagct ctacccgaac gtagacttct acaccggcct gatctaccgc 1140
gcaatgggct tcccaactga cttcttcacc gtattgttcg caatcggtcg tctgccagga 1200
tggatcgctc actaccgcga gcagctcggt gcagcaggca acaagatcaa ccgcccacgc 1260
caggtctaca ccggcaacga atcccgcaag ttggttcctc gcgaggagcg ctaa 1314
<210> 43
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> gltA M312I Up F
<400> 43
gtgaattcga gctcggtacc cgcgggaatc ctgcgttacc gc 42
<210> 44
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> gltA M312I Up R
<400> 44
tgtaaacgcg gtgtccgaag ccgatgaggc ggacgccgtc tt 42
<210> 45
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> gltA M312I Down F
<400> 45
aagacggcgt ccgcctcatc ggcttcggac accgcgttta ca 42
<210> 46
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> gltA M312I Down R
<400> 46
ggtcgactct agaggatccc cttagcgctc ctcgcgagga ac 42
<210> 47
<211> 436
<212> PRT
<213> Unknown
<220>
<223> ilvA_WT
<400> 47
Met Ser Glu Thr Tyr Val Ser Glu Lys Ser Pro Gly Val Met Ala Ser
1 5 10 15
Gly Ala Glu Leu Ile Arg Ala Ala Asp Ile Gln Thr Ala Gln Ala Arg
20 25 30
Ile Ser Ser Val Ile Ala Pro Thr Pro Leu Gln Tyr Cys Pro Arg Leu
35 40 45
Ser Glu Glu Thr Gly Ala Glu Ile Tyr Leu Lys Arg Glu Asp Leu Gln
50 55 60
Asp Val Arg Ser Tyr Lys Ile Arg Gly Ala Leu Asn Ser Gly Ala Gln
65 70 75 80
Leu Thr Gln Glu Gln Arg Asp Ala Gly Ile Val Ala Ala Ser Ala Gly
85 90 95
Asn His Ala Gln Gly Val Ala Tyr Val Cys Lys Ser Leu Gly Val Gln
100 105 110
Gly Arg Ile Tyr Val Pro Val Gln Thr Pro Lys Gln Lys Arg Asp Arg
115 120 125
Ile Met Val His Gly Gly Glu Phe Val Ser Leu Val Val Thr Gly Asn
130 135 140
Asn Phe Asp Glu Ala Ser Ala Ala Ala His Glu Asp Ala Glu Arg Thr
145 150 155 160
Gly Ala Thr Leu Ile Glu Pro Phe Asp Ala Arg Asn Thr Val Ile Gly
165 170 175
Gln Gly Thr Val Ala Ala Glu Ile Leu Ser Gln Leu Thr Ser Met Gly
180 185 190
Lys Ser Ala Asp His Val Met Val Pro Val Gly Gly Gly Gly Leu Leu
195 200 205
Ala Gly Val Val Ser Tyr Met Ala Asp Met Ala Pro Arg Thr Ala Ile
210 215 220
Val Gly Ile Glu Pro Ala Gly Ala Ala Ser Met Gln Ala Ala Leu His
225 230 235 240
Asn Gly Gly Pro Ile Thr Leu Glu Thr Val Asp Pro Phe Val Asp Gly
245 250 255
Ala Ala Val Lys Arg Val Gly Asp Leu Asn Tyr Thr Ile Val Glu Lys
260 265 270
Asn Gln Gly Arg Val His Met Met Ser Ala Thr Glu Gly Ala Val Cys
275 280 285
Thr Glu Met Leu Asp Leu Tyr Gln Asn Glu Gly Ile Ile Ala Glu Pro
290 295 300
Ala Gly Ala Leu Ser Ile Ala Gly Leu Lys Glu Met Ser Phe Ala Pro
305 310 315 320
Gly Ser Val Val Val Cys Ile Ile Ser Gly Gly Asn Asn Asp Val Leu
325 330 335
Arg Tyr Ala Glu Ile Ala Glu Arg Ser Leu Val His Arg Gly Leu Lys
340 345 350
His Tyr Phe Leu Val Asn Phe Pro Gln Lys Pro Gly Gln Leu Arg His
355 360 365
Phe Leu Glu Asp Ile Leu Gly Pro Asp Asp Asp Ile Thr Leu Phe Glu
370 375 380
Tyr Leu Lys Arg Asn Asn Arg Glu Thr Gly Thr Ala Leu Val Gly Ile
385 390 395 400
His Leu Ser Glu Ala Ser Gly Leu Asp Ser Leu Leu Glu Arg Met Glu
405 410 415
Glu Ser Ala Ile Asp Ser Arg Arg Leu Glu Pro Gly Thr Pro Glu Tyr
420 425 430
Glu Tyr Leu Thr
435
<210> 48
<211> 1311
<212> DNA
<213> Unknown
<220>
<223> ilvA_WT_nt
<400> 48
atgagtgaaa catacgtgtc tgagaaaagt ccaggagtga tggctagcgg agcggagctg 60
attcgtgccg ccgacattca aacggcgcag gcacgaattt cctccgtcat tgcaccaact 120
ccattgcagt attgccctcg tctttctgag gaaaccggag cggaaatcta ccttaagcgt 180
gaggatctgc aggatgttcg ttcctacaag atccgcggtg cgctgaactc tggagcgcag 240
ctcacccaag agcagcgcga tgcaggtatc gttgccgcat ctgcaggtaa ccatgcccag 300
ggcgtggcct atgtgtgcaa gtccttgggc gttcagggac gcatctatgt tcctgtgcag 360
actccaaagc aaaagcgtga ccgcatcatg gttcacggcg gagagtttgt ctccttggtg 420
gtcactggca ataacttcga cgaagcatcg gctgcagcgc atgaagatgc agagcgcacc 480
ggcgcaacgc tgatcgagcc tttcgatgct cgcaacaccg tcatcggtca gggcaccgtg 540
gctgctgaga tcttgtcgca gctgacttcc atgggcaaga gtgcagatca cgtgatggtt 600
ccagtcggcg gtggcggact tcttgcaggt gtggtcagct acatggctga tatggcacct 660
cgcactgcga tcgttggtat cgaaccagcg ggagcagcat ccatgcaggc tgcattgcac 720
aatggtggac caatcacttt ggagactgtt gatccctttg tggacggcgc agcagtcaaa 780
cgtgtcggag atctcaacta caccatcgtg gagaagaacc agggtcgcgt gcacatgatg 840
agcgcgaccg agggcgctgt gtgtactgag atgctcgatc tttaccaaaa cgaaggcatc 900
atcgcggagc ctgctggcgc gctgtctatc gctgggttga aggaaatgtc ctttgcacct 960
ggttctgtcg tggtgtgcat catctctggt ggcaacaacg atgtgctgcg ttatgcggaa 1020
atcgctgagc gctccttggt gcaccgcggt ttgaagcact acttcttggt gaacttcccg 1080
caaaagcctg gtcagttgcg tcacttcctg gaagatatcc tgggaccgga tgatgacatc 1140
acgctgtttg agtacctcaa gcgcaacaac cgtgagaccg gtactgcgtt ggtgggtatt 1200
cacttgagtg aagcatcagg attggattct ttgctggaac gtatggagga atcggcaatt 1260
gattcccgtc gcctcgagcc gggcacccct gagtacgaat acttgaccta a 1311
<210> 49
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 49
ggatccgact gagcctgggc aactgg 26
<210> 50
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 50
ggatccccgt caccgacacc tccaca 26
<210> 51
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 51
acatcacgct ggcagagtac ctcaa 25
<210> 52
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 52
ttgaggtact ctgccagcgt gatgt 25
<210> 53
<211> 436
<212> PRT
<213> Artificial Sequence
<220>
<223> ilvA_T381A+F383A
<400> 53
Met Ser Glu Thr Tyr Val Ser Glu Lys Ser Pro Gly Val Met Ala Ser
1 5 10 15
Gly Ala Glu Leu Ile Arg Ala Ala Asp Ile Gln Thr Ala Gln Ala Arg
20 25 30
Ile Ser Ser Val Ile Ala Pro Thr Pro Leu Gln Tyr Cys Pro Arg Leu
35 40 45
Ser Glu Glu Thr Gly Ala Glu Ile Tyr Leu Lys Arg Glu Asp Leu Gln
50 55 60
Asp Val Arg Ser Tyr Lys Ile Arg Gly Ala Leu Asn Ser Gly Ala Gln
65 70 75 80
Leu Thr Gln Glu Gln Arg Asp Ala Gly Ile Val Ala Ala Ser Ala Gly
85 90 95
Asn His Ala Gln Gly Val Ala Tyr Val Cys Lys Ser Leu Gly Val Gln
100 105 110
Gly Arg Ile Tyr Val Pro Val Gln Thr Pro Lys Gln Lys Arg Asp Arg
115 120 125
Ile Met Val His Gly Gly Glu Phe Val Ser Leu Val Val Thr Gly Asn
130 135 140
Asn Phe Asp Glu Ala Ser Ala Ala Ala His Glu Asp Ala Glu Arg Thr
145 150 155 160
Gly Ala Thr Leu Ile Glu Pro Phe Asp Ala Arg Asn Thr Val Ile Gly
165 170 175
Gln Gly Thr Val Ala Ala Glu Ile Leu Ser Gln Leu Thr Ser Met Gly
180 185 190
Lys Ser Ala Asp His Val Met Val Pro Val Gly Gly Gly Gly Leu Leu
195 200 205
Ala Gly Val Val Ser Tyr Met Ala Asp Met Ala Pro Arg Thr Ala Ile
210 215 220
Val Gly Ile Glu Pro Ala Gly Ala Ala Ser Met Gln Ala Ala Leu His
225 230 235 240
Asn Gly Gly Pro Ile Thr Leu Glu Thr Val Asp Pro Phe Val Asp Gly
245 250 255
Ala Ala Val Lys Arg Val Gly Asp Leu Asn Tyr Thr Ile Val Glu Lys
260 265 270
Asn Gln Gly Arg Val His Met Met Ser Ala Thr Glu Gly Ala Val Cys
275 280 285
Thr Glu Met Leu Asp Leu Tyr Gln Asn Glu Gly Ile Ile Ala Glu Pro
290 295 300
Ala Gly Ala Leu Ser Ile Ala Gly Leu Lys Glu Met Ser Phe Ala Pro
305 310 315 320
Gly Ser Val Val Val Cys Ile Ile Ser Gly Gly Asn Asn Asp Val Leu
325 330 335
Arg Tyr Ala Glu Ile Ala Glu Arg Ser Leu Val His Arg Gly Leu Lys
340 345 350
His Tyr Phe Leu Val Asn Phe Pro Gln Lys Pro Gly Gln Leu Arg His
355 360 365
Phe Leu Glu Asp Ile Leu Gly Pro Asp Asp Asp Ile Ala Leu Ala Glu
370 375 380
Tyr Leu Lys Arg Asn Asn Arg Glu Thr Gly Thr Ala Leu Val Gly Ile
385 390 395 400
His Leu Ser Glu Ala Ser Gly Leu Asp Ser Leu Leu Glu Arg Met Glu
405 410 415
Glu Ser Ala Ile Asp Ser Arg Arg Leu Glu Pro Gly Thr Pro Glu Tyr
420 425 430
Glu Tyr Leu Thr
435
<210> 54
<211> 1311
<212> DNA
<213> Artificial Sequence
<220>
<223> ilvA_T381A+F383A_nt
<400> 54
atgagtgaaa catacgtgtc tgagaaaagt ccaggagtga tggctagcgg agcggagctg 60
attcgtgccg ccgacattca aacggcgcag gcacgaattt cctccgtcat tgcaccaact 120
ccattgcagt attgccctcg tctttctgag gaaaccggag cggaaatcta ccttaagcgt 180
gaggatctgc aggatgttcg ttcctacaag atccgcggtg cgctgaactc tggagcgcag 240
ctcacccaag agcagcgcga tgcaggtatc gttgccgcat ctgcaggtaa ccatgcccag 300
ggcgtggcct atgtgtgcaa gtccttgggc gttcagggac gcatctatgt tcctgtgcag 360
actccaaagc aaaagcgtga ccgcatcatg gttcacggcg gagagtttgt ctccttggtg 420
gtcactggca ataacttcga cgaagcatcg gctgcagcgc atgaagatgc agagcgcacc 480
ggcgcaacgc tgatcgagcc tttcgatgct cgcaacaccg tcatcggtca gggcaccgtg 540
gctgctgaga tcttgtcgca gctgacttcc atgggcaaga gtgcagatca cgtgatggtt 600
ccagtcggcg gtggcggact tcttgcaggt gtggtcagct acatggctga tatggcacct 660
cgcactgcga tcgttggtat cgaaccagcg ggagcagcat ccatgcaggc tgcattgcac 720
aatggtggac caatcacttt ggagactgtt gatccctttg tggacggcgc agcagtcaaa 780
cgtgtcggag atctcaacta caccatcgtg gagaagaacc agggtcgcgt gcacatgatg 840
agcgcgaccg agggcgctgt gtgtactgag atgctcgatc tttaccaaaa cgaaggcatc 900
atcgcggagc ctgctggcgc gctgtctatc gctgggttga aggaaatgtc ctttgcacct 960
ggttctgtcg tggtgtgcat catctctggt ggcaacaacg atgtgctgcg ttatgcggaa 1020
atcgctgagc gctccttggt gcaccgcggt ttgaagcact acttcttggt gaacttcccg 1080
caaaagcctg gtcagttgcg tcacttcctg gaagatatcc tgggaccgga tgatgacatc 1140
gcgctggcag agtacctcaa gcgcaacaac cgtgagaccg gtactgcgtt ggtgggtatt 1200
cacttgagtg aagcatcagg attggattct ttgctggaac gtatggagga atcggcaatt 1260
gattcccgtc gcctcgagcc gggcacccct gagtacgaat acttgaccta a 1311
<210> 55
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 55
aacacgaccg gcatcccgtc gc 22
<210> 56
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 56
aaatcatttg agaaaactcg agg 23
<210> 57
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 57
gtgaattcga gctcggtacc caaatcattt gagaaaactc gaggc 45
<210> 58
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 58
ggtgatcatc tcaacggtgg aacacaggtt gatgatcatt gggtt 45
<210> 59
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 59
aacccaatga tcatcaacct gtgttccacc gttgagatga tcacc 45
<210> 60
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 60
ggtcgactct agaggatccc caagaaggca acatcggaca gc 42
<210> 61
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 61
atccattcaa tggagtctgc g 21
<210> 62
<211> 616
<212> PRT
<213> Artificial Sequence
<220>
<223> leuA_P247C+R558H+G561D
<400> 62
Met Ser Pro Asn Asp Ala Phe Ile Ser Ala Pro Ala Lys Ile Glu Thr
1 5 10 15
Pro Val Gly Pro Arg Asn Glu Gly Gln Pro Ala Trp Asn Lys Gln Arg
20 25 30
Gly Ser Ser Met Pro Val Asn Arg Tyr Met Pro Phe Glu Val Glu Val
35 40 45
Glu Asp Ile Ser Leu Pro Asp Arg Thr Trp Pro Asp Lys Lys Ile Thr
50 55 60
Val Ala Pro Gln Trp Cys Ala Val Asp Leu Arg Asp Gly Asn Gln Ala
65 70 75 80
Leu Ile Asp Pro Met Ser Pro Glu Arg Lys Arg Arg Met Phe Glu Leu
85 90 95
Leu Val Gln Met Gly Phe Lys Glu Ile Glu Val Gly Phe Pro Ser Ala
100 105 110
Ser Gln Thr Asp Phe Asp Phe Val Arg Glu Ile Ile Glu Lys Gly Met
115 120 125
Ile Pro Asp Asp Val Thr Ile Gln Val Leu Val Gln Ala Arg Glu His
130 135 140
Leu Ile Arg Arg Thr Phe Glu Ala Cys Glu Gly Ala Lys Asn Val Ile
145 150 155 160
Val His Phe Tyr Asn Ser Thr Ser Ile Leu Gln Arg Asn Val Val Phe
165 170 175
Arg Met Asp Lys Val Gln Val Lys Lys Leu Ala Thr Asp Ala Ala Glu
180 185 190
Leu Ile Lys Thr Ile Ala Gln Asp Tyr Pro Asp Thr Asn Trp Arg Trp
195 200 205
Gln Tyr Ser Pro Glu Ser Phe Thr Gly Thr Glu Val Glu Tyr Ala Lys
210 215 220
Glu Val Val Asp Ala Val Val Glu Val Met Asp Pro Thr Pro Glu Asn
225 230 235 240
Pro Met Ile Ile Asn Leu Cys Ser Thr Val Glu Met Ile Thr Pro Asn
245 250 255
Val Tyr Ala Asp Ser Ile Glu Trp Met His Arg Asn Leu Asn Arg Arg
260 265 270
Asp Ser Ile Ile Leu Ser Leu His Pro His Asn Asp Arg Gly Thr Gly
275 280 285
Val Gly Ala Ala Glu Leu Gly Tyr Met Ala Gly Ala Asp Arg Ile Glu
290 295 300
Gly Cys Leu Phe Gly Asn Gly Glu Arg Thr Gly Asn Val Cys Leu Val
305 310 315 320
Thr Leu Ala Leu Asn Met Leu Thr Gln Gly Val Asp Pro Gln Leu Asp
325 330 335
Phe Thr Asp Ile Arg Gln Ile Arg Ser Thr Val Glu Tyr Cys Asn Gln
340 345 350
Leu Arg Val Pro Glu Arg His Pro Tyr Gly Gly Asp Leu Val Phe Thr
355 360 365
Ala Phe Ser Gly Ser His Gln Asp Ala Val Asn Lys Gly Leu Asp Ala
370 375 380
Met Ala Ala Lys Val Gln Pro Gly Ala Ser Ser Thr Glu Val Ser Trp
385 390 395 400
Glu Gln Leu Arg Asp Thr Glu Trp Glu Val Pro Tyr Leu Pro Ile Asp
405 410 415
Pro Lys Asp Val Gly Arg Asp Tyr Glu Ala Val Ile Arg Val Asn Ser
420 425 430
Gln Ser Gly Lys Gly Gly Val Ala Tyr Ile Met Lys Thr Asp His Gly
435 440 445
Leu Gln Ile Pro Arg Ser Met Gln Val Glu Phe Ser Thr Val Val Gln
450 455 460
Asn Val Thr Asp Ala Glu Gly Gly Glu Val Asn Ser Lys Ala Met Trp
465 470 475 480
Asp Ile Phe Ala Thr Glu Tyr Leu Glu Arg Thr Ala Pro Val Glu Gln
485 490 495
Ile Ala Leu Arg Val Glu Asn Ala Gln Thr Glu Asn Glu Asp Ala Ser
500 505 510
Ile Thr Ala Glu Leu Ile His Asn Gly Lys Asp Val Thr Val Asp Gly
515 520 525
Arg Gly Asn Gly Pro Leu Ala Ala Tyr Ala Asn Ala Leu Glu Lys Leu
530 535 540
Gly Ile Asp Val Glu Ile Gln Glu Tyr Asn Gln His Ala His Thr Ser
545 550 555 560
Asp Asp Asp Ala Glu Ala Ala Ala Tyr Val Leu Ala Glu Val Asn Gly
565 570 575
Arg Lys Val Trp Gly Val Gly Ile Ala Gly Ser Ile Thr Tyr Ala Ser
580 585 590
Leu Lys Ala Val Thr Ser Ala Val Asn Arg Ala Leu Asp Val Asn His
595 600 605
Glu Ala Val Leu Ala Gly Gly Val
610 615
<210> 63
<211> 1851
<212> DNA
<213> Artificial Sequence
<220>
<223> leuA_P247C+R558H+G561D_nt
<400> 63
atgtctccta acgatgcatt catctccgca cctgccaaga tcgaaacccc agttgggcct 60
cgcaacgaag gccagccagc atggaataag cagcgtggct cctcaatgcc agttaaccgc 120
tacatgcctt tcgaggttga ggtagaagat atttctctgc cggaccgcac ttggccagat 180
aaaaaaatca ccgttgcacc tcagtggtgt gctgttgacc tgcgtgacgg caaccaggct 240
ctgattgatc cgatgtctcc tgagcgtaag cgccgcatgt ttgagctgct ggttcagatg 300
ggcttcaaag aaatcgaggt cggtttccct tcagcttccc agactgattt tgatttcgtt 360
cgtgagatca tcgaaaaggg catgatccct gacgatgtca ccattcaggt tctggttcag 420
gctcgtgagc acctgattcg ccgtactttt gaagcttgcg aaggcgcaaa aaacgttatc 480
gtgcacttct acaactccac ctccatcctg cagcgcaacg tggtgttccg catggacaag 540
gtgcaggtga agaagctggc taccgatgcc gctgaactaa tcaagaccat cgctcaggat 600
tacccagaca ccaactggcg ctggcagtac tcccctgagt ccttcaccgg cactgaggtt 660
gagtacgcca aggaagttgt ggacgcagtt gttgaggtca tggatccaac tcctgagaac 720
ccaatgatca tcaacctgtg ttccaccgtt gagatgatca cccctaacgt ttacgcagac 780
tccattgaat ggatgcaccg caatctaaac cgtcgtgatt ccattatcct gtccctgcac 840
ccgcacaatg accgtggcac cggcgttggc gcagctgagc tgggctacat ggctggcgct 900
gaccgcatcg aaggctgcct gttcggcaac ggcgagcgca ccggcaacgt ctgcctggtc 960
accctggcac tgaacatgct gacccagggc gttgaccctc agctggactt caccgatata 1020
cgccagatcc gcagcaccgt tgaatactgc aaccagctgc gcgttcctga gcgccaccca 1080
tacggcggtg acctggtctt caccgctttc tccggttccc accaggacgc tgtgaacaag 1140
ggtctggacg ccatggctgc caaggttcag ccaggtgcta gctccactga agtttcttgg 1200
gagcagctgc gcgacaccga atgggaggtt ccttacctgc ctatcgatcc aaaggatgtc 1260
ggtcgcgact acgaggctgt tatccgcgtg aactcccagt ccggcaaggg cggcgttgct 1320
tacatcatga agaccgatca cggtctgcag atccctcgct ccatgcaggt tgagttctcc 1380
accgttgtcc agaacgtcac cgacgctgag ggcggcgagg tcaactccaa ggcaatgtgg 1440
gatatcttcg ccaccgagta cctggagcgc accgcaccag ttgagcagat cgcgctgcgc 1500
gtcgagaacg ctcagaccga aaacgaggat gcatccatca ccgccgagct catccacaac 1560
ggcaaggacg tcaccgtcga tggccgcggc aacggcccac tggccgctta cgccaacgcg 1620
ctggagaagc tgggcatcga cgttgagatc caggaataca accagcacgc ccacacctcg 1680
gatgacgatg cagaagcagc cgcctacgtg ctggctgagg tcaacggccg caaggtctgg 1740
ggcgtcggca tcgctggctc catcacctac gcttcgctga aggcagtgac ctccgccgta 1800
aaccgcgcgc tggacgtcaa ccacgaggca gtcctggctg gcggcgttta a 1
851
<---
Изобретение относится к биотехнологии. Предложен полипептид, вовлеченный в получение аминокислоты с разветвленной цепью, в котором аминокислота в любом одном или более чем одном соответствующем положении 217, 310, 403 и 462 от N-конца в аминокислотной последовательности SEQ ID NO: 1 заменена на другую аминокислоту, где другая аминокислота представляет собой аланин (Ala), где полипептид обладает гомологией или идентичностью 90% или более с SEQ ID NO: 1. Также предложены полинуклеотид, кодирующий указанный полипептид, экспрессионный вектор, содержащий указанный полинуклеотид, микроорганизм рода Corynebacterium, продуцирующий аминокислоту с разветвленной цепью и содержащий одно или более чем одно из следующего: указанные полипептид, полинуклеотид и вектор. Также предложен способ получения аминокислоты с разветвленной цепью. Изобретение обеспечивает получение аминокислоты с разветвленной цепью с высоким выходом и уменьшение выхода побочных продуктов. 5 н. и 5 з.п. ф-лы, 19 табл., 6 пр.
1. Полипептид, вовлеченный в получение аминокислоты с разветвленной цепью, в котором аминокислота в любом одном или более чем одном соответствующем положении 217, 310, 403 и 462 от N-конца в аминокислотной последовательности SEQ ID NO: 1 заменена на другую аминокислоту, где другая аминокислота представляет собой аланин (Ala), где полипептид обладает гомологией или идентичностью 90% или более с SEQ ID NO: 1.
2. Полипептид по п. 1, где полипептид обладает гомологией или идентичностью 99% или более с SEQ ID NO: 3, SEQ ID NO: 5, SEQ ID NO: 7, SEQ ID NO: 9, SEQ ID NO: 23 или SEQ ID NO: 25.
3. Полипептид по п. 1, где полипептид обладает ослабленной активностью по сравнению с полипептидом в соответствии с SEQ ID NO: 1.
4. Полинуклеотид, кодирующий полипептид, по любому из пп. 1-3.
5. Экспрессионный вектор, содержащий полинуклеотид по п. 4.
6. Микроорганизм рода Corynebacterium, продуцирующий аминокислоту с разветвленной цепью, содержащий одно или более чем одно из следующего: полипептид по любому из пп. 1-3, полинуклеотид, кодирующий указанный полипептид; и вектор, содержащий указанный полинуклеотид.
7. Микроорганизм по п. 6, где микроорганизм рода Corynebacterium представляет собой Corynebacterium glutamicum.
8. Способ получения аминокислоты с разветвленной цепью, включающий культивирование микроорганизма по п. 6 в среде.
9. Способ по п. 8, дополнительно включающий выделение аминокислоты с разветвленной цепью из микроорганизма или среды.
10. Способ по п. 8, где аминокислота с разветвленной цепью выбрана из L-лейцина, L-изолейцина и L-валина.
| База данных UniProtKB, A0A1B4WMM4_CORGT, 02.11.2016 | |||
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| Найдено онлайн: https://www.uniprot.org/uniprotkb/A0A1B4WMM4/entry Дата обращения 05.03.2024 | |||
| JP 5236947 A, 17.09.1993 | |||
| СПОСОБ ПОЛУЧЕНИЯ СВЕРХТВЕРДОГО КОМПОЗИЦИОННОГО МАТЕРИАЛА НА ОСНОВЕ КУБИЧЕСКОГО НИТРИДА БОРА ДЛЯ РЕЖУЩИХ ИНСТРУМЕНТОВ И КОМПОЗИЦИОННЫЙ МАТЕРИАЛ | 1999 |
|
RU2147972C1 |
| 3-ДЕЗОКСИ-D-АРАБИНОГЕПТУЛОЗОНАТ-7-ФОСФАТСИНТАЗА И ФРАГМЕНТ ДНК, КОДИРУЮЩИЙ 3-ДЕЗОКСИ-D-АРАБИНОГЕПТУЛОЗОНАТ-7-ФОСФАТСИНТАЗУ ИЗ METHYLOPHILUS METHYLOTROPHUS | 2000 |
|
RU2229514C2 |
Авторы
Даты
2024-09-11—Публикация
2021-11-18—Подача