Область техники
Изобретение относится к биотехнологии и может быть использовано для получения белка RBD (с англ.: receptor-binding-domain) – рекомбинантного структурного домена гликопротеина spike сарбековируса SARS-CoV-2 линии B.1.1.529. Получаемый белок RBD предназначен для непосредственного производства вакцинного препарата против SARS-CoV-2 линии B.1.1.529(Yang et al., 2020; Min, Sun 2021). Способ включает дизайн генетических конструкций, методы трансфекции клеточных линий, очистки и оценки экспрессии целевого белка.
Уровень техники
Рецептор-связывающий домен RBD является частью субъединицы S1 белка S (spike). Использование рекомбинантного структурного домена RBD в качестве антигена при производстве вакцинных препаратов против SARS-CoV-2 имеет ряд преимуществ, связанных с выработкой большого числа нейтрализующих антител в ответ на введение антигена (Zhou et al., 2019; Shang et al., 2020; Song et al., 2018; Yang et al., 2020; Min, Sun 2021; Yang, Du, 2011), что было продемонстрировано рядом научных исследований, а также высокой консервативностью региона. На данный момент известно 13 кандидатных вакцин, основанных на использовании домена RBD (https://www.who.int/publications/m/item/draft-landscape-of-covid-19-candidate-vaccines).
В связи с высоким молекулярно-генетическим полиморфизмом вируса SARS-CoV-2 и появлением новых линий с мутациями, ассоциированными с повышенной трансмиссивностью и тяжестью течения инфекционного заболевания (https://www.ecdc.europa.eu/en/covid-19/variants-concern; https://www.gisaid.org), а также вероятностью снижения эффективности ранее разработанных вакцин, в изобретении использован вариант антигена RBD линии B.1.1.529коронавируса SARS-CoV-2.
Известен патент «Способ получения штамма клеток яичника китайского хомячка, продуцента рекомбинантного белка RBD вируса SARS-CoV-2, штамм клеток яичника китайского хомячка, продуцент рекомбинантного белка RBD вируса SARS-CoV-2, способ получения рекомбинантного белка RBD вируса SARS-CoV-2, тест-система для иммуноферментного анализа сыворотки или плазмы крови человека и ее применение» (RU2723008C9, https://patents.google.com/patent/RU2723008C9), однако основным назначением получаемого рекомбинантного белка – применение для диагностических целей, а не для использования в составе вакцинных препаратов. К недостаткам данного способа также следует отнести относительно низкий уровень экспрессии целевого белка (до 10 мг/л), что связано, прежде всего, с особенностями проведённого дизайна молекулярно-генетической векторной конструкции.
Наиболее близким по сущности к заявленному изобретению является изобретение «SARS-CoV-2-RBD eucaryotic protein expression vector and its preparation method and use» (CN112831523A, https://patents.google.com/patent/CN112831523A), где для экспрессии белка использован вектор pcDNA3.1, содержащий сигнальный пептид tPA. Экспрессия осуществляется в клетках 293F. Отличием является использование другой экспрессионной системы. К недостаткам данного способа следует отнести используемый дизайн ДНК, кодирующей последовательность RBD, не учитывающий современные представления о молекулярно-генетическом полиморфизме вируса SARS-CoV-2. Также
Также известен патент «SARS-CoV-2N/S1(RBD) recombinant protein and its preparation method and application» (CN111607003A, https://patents.google.com/patent/CN111607003A), в котором приводится способ получения и применение рекомбинантных белков нуклеокапсида (N) и RBD. К недостаткам данного изобретения относится выбранная экспрессионная система (прокариотическая), не позволяющая проводить полноценные посттрансляционные преобразования (фолдинг, гликозилирование и пр.). К недостаткам также следует отнести используемый дизайн фрагмента RBD, не учитывающий современные представления о молекулярно-генетическом полиморфизме вируса SARS-CoV-2.
Раскрытие изобретения
Задачей настоящего изобретения являлась разработка рекомбинантной плазмиды и структуры антигена для получения рекомбинантного белка RBDomik, соответствующего линии B.1.1.529 коронавируса SARS-CoV-2.
Техническим результатом изобретения является получение рекомбинантной плазмиды, содержащей нуклеотидную последовательность домена RBDomicron с оптимизированными кодонами и обеспечивающей экспрессию белка RBD линии B.1.1.529 SARS-CoV-2 в клетках млекопитающих.
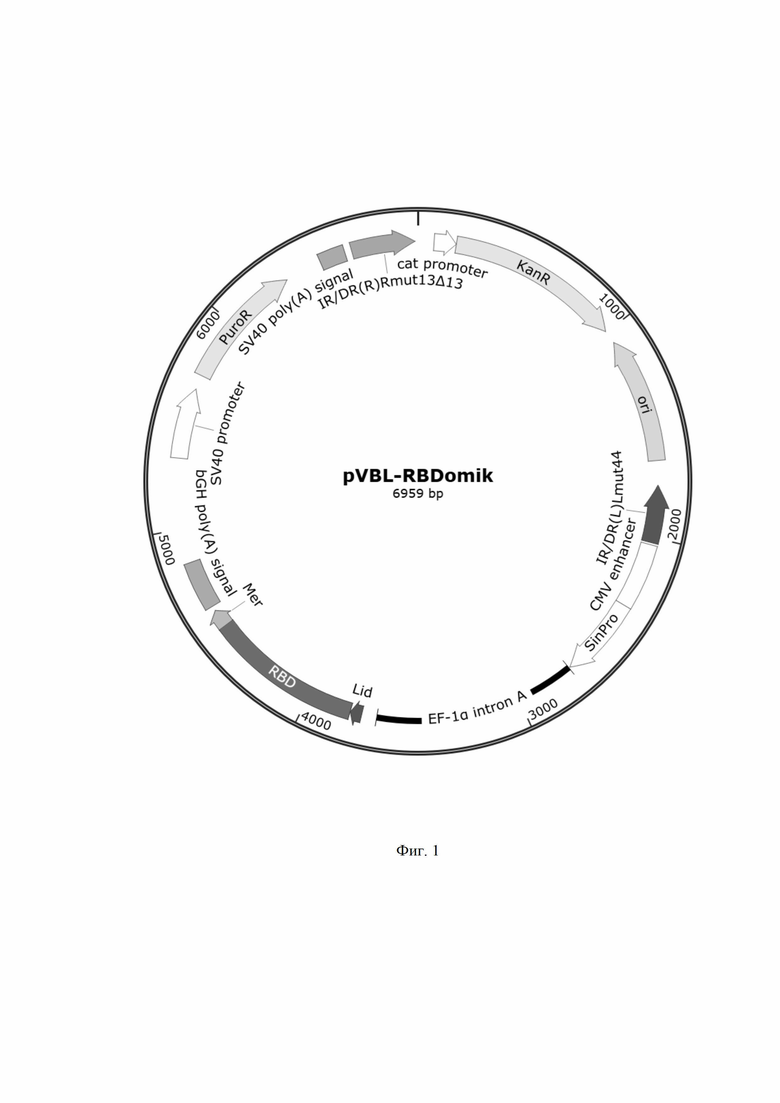
Поставленная техническая проблема решается получением кодон-оптимизированной нуклеотидной последовательности, имеющей последовательность SEQ ID NO: 1, кодирующей белок RBD SARS-CoV-2, а также тем, что получена рекомбинантная плазмида pVBL-RBDomik (фиг. 1), содержащая оптимизированную нуклеотидную последовательность SEQ ID NO:2 и обеспечивающая экспрессию белка RBD в клетках млекопитающих.
Поставленная техническая проблема решается также тем, что разработан способ получения белка RBD с аминокислотной последовательностью SEQ ID NO:3, включающий культивирование клеток эукариот и экспрессию целевого белка, выделение белка с помощью аффинной хроматографии и скрининг экспрессии.
Изобретение имеет ряд преимуществ по сравнению с близкими по сущности аналогами. Использование белка RBD, характерного для линии B.1.1.529 коронавируса SARS-CoV-2, позволит его использовать для создания эффективных вакцинных препаратов, действующих в том числе против высокопатогенной и высококонтагиозной линии B.1.1.529. Проведенная оптимизация кодонов фрагмента ДНК, кодирующего белок RBD, обеспечивает высокий уровень экспрессии белка. Экспрессия белка в эукариотических системах позволяет получать белок с необходимыми посттрансляционными модификациями. Использование в составе продуцируемого белка последовательности HisTag делает возможным оптимизацию способа выделения и очистки белка RBD.
Краткое описание фигур
Рекомбинантная плазмида pVBL-RBDomik проиллюстрирована на фиг. 1, где показаны основные структурно-функциональные регионы:
- cat promoter (76–178п.н.);
- последовательность NeoR/KanR (179-994 п.н.)
- участок начала репликации ori (1057–1644 п.н.);
- CMV энхансер (2035–23п.н.38 п.н.);
- IR/DR-L (1760–2024п.н.);
- SinPro (2339-2721)
- Chimeric intron (2726–3670 п.н.) – предназначен для усиления экспрессии целевого гена;
- лидерный пептид Lid (3742-3795 п.н.), обеспечивающий экспорт белка из клетки;
- нуклеотидная последовательность гена RBD (3796–4497 п.н.);
- последовательность PuroR(5714–6313п.н.);
- SV40 poly(A) signal(6487–6608 п.н.)– необходим для полиаденилирования;
- последовательность IR/DR-R (6642–6937 п.н);
- Mer (4498–4581 п.н.) – необходим для правильной тримеризации и фолдинга белка.
Сведения, подтверждающие возможность осуществления изобретения
Ниже приведены примеры 1–3 конкретного осуществления изобретения.
Осуществление изобретения
Пример 1. Конструирование рекомбинантной плазмиды pVBL-RBDomicron для синтеза RBDomicron SARS-CoV-2 в клетках млекопитающих
Последовательность гена, кодирующего домен RBDomik (308V – 541F) коронавируса SARS-CoV-2 (GenBank OL677199.1) заимствовали из базы данных GenBank и проводили оптимизацию частот встречаемости кодонов для млекопитающих при помощи сервиса JCat (http://www.jcat.de/), при этом индекс адаптации кодонов CAI достиг 0,85.
На основе вектора pVBL получали рекомбинантную плазмиду pVBL-RBDomik (фиг. 1), содержащую нуклеотидную последовательность RBDomik (SEQ ID NO:1)и лидерный пептид Lid белка люциферазы гауссии (MGVKVLFALICIAVAEAS), а также последовательность Mer,которая необходима для правильной тримеризации и фолдинга белка.
Нуклеотидную последовательность сигнального пептида Lid, С-концевой домен фибритина бактериофага Citrobacter Merlin и сайты рестрикции PspCI и EcoICRI вводили в последовательность RBDomik одновременно с помощью ПЦР. Использовались пары праймеров F_RBDomik_ PspCI и R_RBDH_EcoICRI (таблица 1).
Таблица 1 – Нуклеотидные последовательности праймеров

Пример 2. Создание рекомбинантной плазмиды (pVBL-RBDomik).
2.1. Амплификация методом ПЦР нуклеотидной последовательности, кодирующей RBDomikSARS-CoV-2
Амплификацию фрагментов проводили по стандартному протоколу. Реакционная смесь, объемом 50 мкл содержала 10×буфер, 25 мМ dNTP, олигонуклеотидные праймеры (таблица 1) в количестве 10 пмоль каждого, 5 ед. а Pfu-полимеразы, 1 мкл раствора плазмидной ДНК. Реакцию проводили в амплификаторе Veriti™ 96-Well Thermal Cycler. Температурно-временной профиль ПЦР: плавление – 95ºС – 5 мин. (1 цикл). 95ºС – 30 секунд. Отжиг – 58ºС – 30 секунд, элонгация – 72ºС – 30 секунд (30 циклов).
2.2.Выделение ДНК из агарозного геля
После визуализации под УФ-излучением фрагменты ДНК необходимой длины, разделенные в агарозном геле, вырезали из геля и элюировали при помощи набора «Евроген» CleanupStandard (Россия) в соответствии с рекомендациями производителя.
2.3. Ферментативный гидролиз вектора pVBL и ПЦР-продукта (последовательность участка генома SARS-CoV-2, кодирующей RBDomik)
Для клонирования гена RBDomikSARS-CoV-2 в составе вектора pVBL были использованы эндонуклеазы рестрикции PspCI и EcoICRI (фирма «Сибэнзим», Россия) с прилагаемыми к ним буферами. Реакционную смесь готовили в соответствии с активностью фермента (2-5 е.а. на 1 мкг ДНК) и концентрации плазмидной ДНК. Условия реакции: температура, состав буфера и длительность проведения ферментативного гидролиза ДНК подбирали в соответствии с инструкциями производителя “Сибэнзим”.
2.4. Лигирование ПЦР-продукта (последовательность участка генома SARS-CoV-2, кодирующей RBDomik) и вектора pVBL
Реакцию лигирования проводили в течение 30 мин. при комнатной температуре, используя смесь из 2 мкг ампликонов с ДНК-матрицы, 1 мкг векторной плазмиды и 20 е.а. ДНК-лигазы фага Т4 в прилагаемом к коммерческому набору реакционном буфере. Полученную лигазную смесь использовали для трансформирования культуры компетентных клеток E. coli штамм Neb Stable.
2.5 Heat-shock трансформация
К «компетентным» клеткам E.coli штамм Neb Stable добавляли 10 мкл лигазной смеси (отношение 1:10), инкубировали на льду в течение 30 мин. После этого клетки подвергали «температурному шоку» при 42ºС в течение 45 сек. Охлаждали клетки на льду в течение 2 мин, затем добавляли 200 мкл среды SOB и инкубировали при 37ºС в течение 60 мин. По окончании инкубации трансформированные клетки высевали на чашку Петри с твёрдой питательной средой LB, содержащей антибиотик.
2.6 Отбор клонов для рестрикционного анализа
Клетки E. coli штамма Neb Stable, трансформированные ДНК-вакцинной конструкцией селективно культивировали в 10 мл жидкой питательной среды LB с добавлением антибиотика в рабочей концентрации 25 мкг/мл.
2.7 Определение нуклеотидной последовательности ДНК
Секвенирование проводили по методу Сэнгера. Использовался набор CEQ2000Dye Terminator Cycle Sequencing Kit и 16-капилярный автоматический секвенатор ABI 3130xl.
Пример 3. Трансфекция клеточной линии CHO-K1, рекомбинантной плазмидой pVBL-RBDomik
Клетки CHO-K1 культивировали в CO2-инкубаторе при содержании углекислого газа5% и влажности 80%. Трансфекцию проводили при достижении плотности монослоя 80%.
При достижении 80% плотности монослоя проводили трансфекцию клеток рекомбинантной плазмиды pVBL-RBDomik с помощью реагента Lipofectamine 3000 (ThermoFisher).
Пример 4. Получение рекомбинантного белка RBDomik
Клетки CHO-K1, трансфецированные рекомбинантной плазмидой pVBL-RBDomik, культивировали на роллерных установках и собирали культуральную среду. Наличие целевого белка определяли с помощью ДСН-ПААГ и иммуноблоттинга с использованием сывороток, переболевших COVID-19.
Культуральную жидкость центрифугировали, фильтровали с использованием фильтров с диаметром пор 0,22 мкм. Очитку рекомбинантного белка проводили ионообменной хроматографией на соединенных последовательно колонках с катионообменным (SP-сефароза) и анионообменным (Q-сефароза) сорбентами, уравновешенными 20мМ Трис-HCl, рН 8,2. После нанесения белка колонки промывали 20мМ Трис-HCl, рН 8,2. Затем колонку с сорбентом SP-сефароза, на которую RBD не сорбируется в этих условиях (рН 8,2), отсоединяли, а белки, связавшиеся с сорбентом Q-сефароза, элюировали в линейном градиенте концентрации NaCl от 0 до 1М в 20мМ Трис-HCl, рН 8,2.
Полученные фракции анализировали с помощью вертикального гель-электрофореза в денатурирующих условиях, при условии, что целевой белок имеет молекулярный вес около 31,4 кДа. Фракции с целевым белком диализовали против фосфатно-солевого буфера, далее проводили стерилизующую фильтрацию. Концентрация белка, определенная спектрофотометрически, составила 5-10 мг на литр культуральной среды.
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Общество с ограниченной ответственностью «Эс Джи»
<120> Рекомбинантная плазмида pVBL-RBDomik, обеспечивающая синтез и секрецию рекомбинантного рецептор-связывающего домена (RBD) коронавируса SARS-CoV-2 линии B.1.617 в клетках млекопитающих.
<212> RNA
<213> Homo sapiens
<223> Нуклеотидная последовательность RBDomik
<210> 1
<211> 702
<400>
GTGGAAAAGGGCATCTACCAGACCAGCAACTTCCGGGTGCAGCCCACCGA 50
ATCCATCGTGCGGTTCCCCAATATCACCAATCTGTGCCCCTTCGACGAGG 100
TGTTCAATGCCACCAGATTCGCCTCTGTGTACGCCTGGAACCGGAAGCGG 150
ATCAGCAATTGCGTGGCCGACTACTCCGTGCTGTACAACCTGGCCCCCTT 200
CTTCACCTTCAAGTGCTACGGCGTGTCCCCTACCAAGCTGAACGACCTGT 250
GCTTCACAAACGTGTACGCCGACAGCTTCGTGATCCGGGGAGATGAAGTG 300
CGGCAGATTGCCCCTGGACAGACAGGCAACATCGCCGACTACAACTACAA 350
GCTGCCCGACGACTTCACCGGCTGTGTGATTGCCTGGAACAGCAACAAGC 400
TGGACTCCAAAGTCTCCGGCAACTACAATTACCTGTACCGGCTGTTCCGG 450
AAGTCCAATCTGAAGCCCTTCGAGCGGGACATCTCCACCGAGATCTATCA 500
GGCCGGCAACAAGCCTTGTAACGGCGTGGCCGGCTTCAACTGCTACTTCC 550
CACTGAAGTCCTACTCCTTTCGGCCCACATACGGCGTGGGCCACCAGCCC 600
TACAGAGTGGTGGTGCTGAGCTTCGAACTGCTGCATGCCCCTGCCACAGT 650
GTGCGGCCCTAAGAAAAGCACCAATCTCGTGAAGAACAAATGCGTGAACT 700
TCAACTTCAAC
<223> Нуклеотидная последовательность вектора pVBL-RBDomik
<210> 2
<211> 7110
<400> TCAATACTGACCATTTAAATCATACCTGACCTCCATAGCAGAAAGTCAAAAGCCTCCGACCGGAGGCTTTTGACTTGATCGGCACGTAAGAGGTTCCAACTTTCACCATAATGAAATAAGATCACTACCGGGCGTATTTTTTGAGTTATCGAGATTTTCAGGAGCTAAGGAAGCTAAAATGAGCCATATTCAACGGGAAACGTCTTGCTCGAGGCCGCGATTAAATTCCAACATGGATGCTGATTTATATGGGTATAAATGGGCTCGCGATAATGTCGGGCAATCAGGTGCGACAATCTATCGATTGTATGGGAAGCCCGATGCGCCAGAGTTGTTTCTGAAACATGGCAAAGGTAGCGTTGCCAATGATGTTACAGATGAGATGGTCAGGCTAAACTGGCTGACGGAATTTATGCCTCTTCCGACCATCAAGCATTTTATCCGTACTCCTGATGATGCATGGTTACTCACCACTGCGATCCCAGGGAAAACAGCATTCCAGGTATTAGAAGAATATCCTGATTCAGGTGAAAATATTGTTGATGCGCTGGCAGTGTTCCTGCGCCGGTTGCATTCGATTCCTGTTTGTAATTGTCCTTTTAACGGCGATCGCGTATTTCGTCTGGCTCAGGCGCAATCACGAATGAATAACGGTTTGGTTGGTGCGAGTGATTTTGATGACGAGCGTAATGGCTGGCCTGTTGAACAAGTCTGGAAAGAAATGCATAAGCTTTTGCCATTCTCACCGGATTCAGTCGTCACTCATGGTGATTTCTCACTTGATAACCTTATTTTTGACGAGGGGAAATTAATAGGTTGTATTGATGTTGGACGAGTCGGAATCGCAGACCGATACCAGGATCTTGCCATCCTATGGAACTGCCTCGGTGAGTTTTCTCCTTCATTACAGAAACGGCTTTTTCAAAAATATGGTATTGATAATCCTGATATGAATAAATTGCAGTTTCACTTGATGCTCGATGAGTTTTTCTAATGAGGGCCCAAATGTAATCACCTGGCTCACCTTCGGGTGGGCCTTTCTGCGTTGCTGGCGTTTTTCCATAGGCTCCGCCCCCCTGACGAGCATCACAAAAATCGATGCTCAAGTCAGAGGTGGCGAAACCCGACAGGACTATAAAGATACCAGGCGTTTCCCCCTGGAAGCTCCCTCGTGCGCTCTCCTGTTCCGACCCTGCCGCTTACCGGATACCTGTCCGCCTTTCTCCCTTCGGGAAGCGTGGCGCTTTCTCATAGCTCACGCTGTAGGTATCTCAGTTCGGTGTAGGTCGTTCGCTCCAAGCTGGGCTGTGTGCACGAACCCCCCGTTCAGCCCGACCGCTGCGCCTTATCCGGTAACTATCGTCTTGAGTCCAACCCGGTAAGACACGACTTATCGCCACTGGCAGCAGCCACTGGTAACAGGATTAGCAGAGCGAGGTATGTAGGCGGTGCTACAGAGTTCTTGAAGTGGTGGCCTAACTACGGCTACACTAGAAGAACAGTATTTGGTATCTGCGCTCTGCTGAAGCCAGTTACCTCGGAAAAAGAGTTGGTAGCTCTTGATCCGGCAAACAAACCACCGCTGGTAGCGGTGGTTTTTTTGTTTGCAAGCAGCAGATTACGCGCAGAAAAAAAGGATCTCAAGAAGATCCTTTGATTTTCTACCGAAGAAAGGCCCACCCGTGAAGGTGAGCCAGTGAGTTGATTGCAGTCCAGTTACGCTGGAGTCTGAGGCTCGTCCTGAATGGATGGAAGTTTACATACACTTAAGTTGGAGTCATTAAAACTCGTTTTTCAACTACTCCACAAATTTCTTGTTAACAAACAATAGTTTTGGCAAGTCAGTTAGGACATCTACTTTGTGCATGACACAAGTCATTTTTCCAACAATTGTTTACAGACAGATTATTTCACTTATAATTCACTGTATCACAATTCCAGTGGGTCAGAAGTTTACATACACTAAGTTGACTGTGCCTTTAAACAGCTTGGAAAATTCCAGAAAATGATGTCATGGCTTTAGAAGCCTGCAGGCGTTACATAACTTACGGTAAATGGCCCGCCTGGCTGACCGCCCAACGACCCCCGCCCATTGACGTCAATAATGACGTATGTTCCCATAGTAACGCCAATAGGGACTTTCCATTGACGTCAATGGGTGGAGTATTTACGGTAAACTGCCCACTTGGCAGTACATCAAGTGTATCATATGCCAAGTACGCCCCCTATTGACGTCAATGACGGTAAATGGCCCGCCTGGCATTATGCCCAGTACATGACCTTATGGGACTTTCCTACTTGGCAGTACATCTACGTATTAGTCATCGCTATTACCATGATGATGCGGTTTTGGCAGTACATCAATGGGCGTGGATAGCGGTTTGACTCACGGGGATTTCCAAGTCTCCACCCCATTGACGTCAATGGGAGTTTGTTTTGACTAGTCAGGGCCCCAACCCCCCCAAGCCCCCATTTCACAACACGCTGGCGCTACAGGCGCGTGACTTCCCCTTGCTTTGGGGCGGGGGGCTGAGACTCCTATGTGCTCCGGATTGGTCAGGCACGGCCTTCGGCCCCGCCTCCTGCCACCGCAGATTGGCCGCTAGGCCTCCCCGAGCGCCCTGCCTCCGAGGGCCGGCGCACCATAAAAGAAGCCGCCCTAGCCACGTCCCCTCGCAGTTCGGCGGTCCCGCGGGTCTGTCTCAAGCTTGCCGCCAGAACACAGGTAAGTGCCGTGTGTGGTTCCCGCGGGCCTGGCCTCTTTACGGGTTATGGCCCTTGCGTGCCTTGAATTACTTCCATGCCCCTGGCTGCAGTACGTGATTCTTGATCCCGAGCTTCGGGTTGGAAGTGGGTGGGAGAGTTCGAGGCCTTGCGCTTAAGGAGCCCCTTCGCCTCGTGCTTGAGTTGAGGCCTGGCTTGGGCGCTGGGGCCGCCGCGTGCTAATCTGGTGGCACCTTCGCGCCTGTCTCGCTGCTTTCGCTAAGTCTCTAGCCATTTAAAATTTTTGATAACCAGCTGCGACGCTTTTTTTCTGGCGAGATAGTCTTGTAAATGCGGGCCAAGATCTGCACACTGGTATTTCGGTTTTTGGGGCCGCGGGCGGCGACGGGGCCCGTGCGTCCCAGCGCACATGTTCGGCGAGGCGGGGCCTGCGAGCGCGGCCACCGAGAATCGGACGGGGGTAGTCTCAAACTGGCCGGCCTGCTCTGGTGCCTGGCCTCGCGCCGCCGTGTATCGCCCCGCCCTGGGCGGCAAGGCTGGCCCGGTCGGCACCAGTTGCGTGAGCGGAAAGATGGCCGCTTCCCGGCCCTGCTGCAGGGAGCTCAAAATGGAGGACGCGGCGCCCGGGAGAGCGGGCGGGTGAGTCACCCACACAAAGGAAAAGGGCCTTTCCTTCCTCATCCGTCGCTTCATGTGACTCCACGGAGTACCGGGCGCCGTCCAGGCACCTCGATTAGTTCTCGAGCTTTTGGAGTACGTCGTCTTTAGGTTGGGGGGAGGGGTTTTATGCGATGGAGTTTCCCCACACTGAGTGGGTGGAGACTGAAGAGTTAGGCCAGCTTGGCACTTGATGTAATTCTCCTTGGAATTTGCCCTTTTTGAGTTTGGATCTTGCCTCATTCTCAAGCCTCAGACAGTGGTTCAAAGTTTTTTTCTTCCATTTCAGCTCCTGGGCAACGTTAGCCTCGAGAATTCGTCCTGCTGCGCACGTGGGAAGCCCTGGCCCCGGCCGCCACCATGGGAGTGAAGGTGCTGTTCGCCCTGATCTGTATTGCCGTGGCCGAAGCTTCTGTGGAAAAGGGCATCTACCAGACCAGCAACTTCCGGGTGCAGCCCACCGAATCCATCGTGCGGTTCCCCAATATCACCAATCTGTGCCCCTTCGACGAGGTGTTCAATGCCACCAGATTCGCCTCTGTGTACGCCTGGAACCGGAAGCGGATCAGCAATTGCGTGGCCGACTACTCCGTGCTGTACAACCTGGCCCCCTTCTTCACCTTCAAGTGCTACGGCGTGTCCCCTACCAAGCTGAACGACCTGTGCTTCACAAACGTGTACGCCGACAGCTTCGTGATCCGGGGAGATGAAGTGCGGCAGATTGCCCCTGGACAGACAGGCAACATCGCCGACTACAACTACAAGCTGCCCGACGACTTCACCGGCTGTGTGATTGCCTGGAACAGCAACAAGCTGGACTCCAAAGTCTCCGGCAACTACAATTACCTGTACCGGCTGTTCCGGAAGTCCAATCTGAAGCCCTTCGAGCGGGACATCTCCACCGAGATCTATCAGGCCGGCAACAAGCCTTGTAACGGCGTGGCCGGCTTCAACTGCTACTTCCCACTGAAGTCCTACTCCTTTCGGCCCACATACGGCGTGGGCCACCAGCCCTACAGAGTGGTGGTGCTGAGCTTCGAACTGCTGCATGCCCCTGCCACAGTGTGCGGCCCTAAGAAAAGCACCAATCTCGTGAAGAACAAATGCGTGAACTTCAACTTCAACACCTACATTCCTGAGGCTCCCAGAGATGGCAAGGCCTACGTCAGAAAGGATGGCGCTTGGGTCGACCTGGAAACCCTGTTGCCTTAAGTCGACCGAGCTCAGCCTCGACTGTGCCTTCTAGTTGCCAGCCATCTGTTGTTTGCCCCTCCCCCGTGCCTTCCTTGACCCTGGAAGGTGCCACTCCCACTGTCCTTTCCTAATAAAATGAGGAAATTGCATCGCATTGTCTGAGTAGGTGTCATTCTATTCTGGGGGGTGGGGTGGGGCAGGACAGCAAGGGGGAGGATTGGGAAGACAATAGCAGGCATGCTGGGGATGCGGTGGGCTCTATGGCTTCTGAGGCGGAAAGAACCAGCTGGGGCTCTAGGGGGTATCCCCACGCGCCCTGTAGCGGCGCATTAAGCGCGGCGGGTGTGGTGGTTACGCGCAGCGTGACCGCTACACTTGCCAGCGCCCTAGCGCCCGCTCCTTTCGCTTTCTTCCCTTCCTTTCTCGCCACGTTCGCCGGCTTTCCCCGTCAAGCTCTAAATCGGGGGCTCCCTTTAGGGTTCCGATTTAGTGCTTTACGGCACCTCGACCCCAAAAAACTTGATTAGGGTGATGGTTCACGTAGTGGGCCATCGCCCTGATAGACGGTTTTTCGCCCTTTGACGTTGGAGTCCACGTTCTTTAATAGTGGACTCTTGTTCCAAACTGGAACAACACTCAACCCTATCTCGGTCTATTCTTTTGATTTATAAGGGATTTTGCCGATTTCGGCCTATTGGTTAAAAAATGAGCTGATTTAACAAAAATTTAACGCGAATTAATTCTGTGGAATGTGTGTCAGTTAGGGTGTGGAAAGTCCCCAGGCTCCCCAGCAGGCAGAAGTATGCAAAGCATGCATCTCAATTAGTCAGCAACCAGGTGTGGAAAGTCCCCAGGCTCCCCAGCAGGCAGAAGTATGCAAAGCATGCATCTCAATTAGTCAGCAACCATAGTCCCGCCCCTAACTCCGCCCATCCCGCCCCTAACTCCGCCCAGTTCCGCCCATTCTCCGCCCCATGGCTGACTAATTTTTTTTATTTATGCAGAGGCCGAGGCCGCCTCTGCCTCTGAGCTATTCCAGAAGTAGTGAGGAGGCTTTTTTGGAGGCCTAGGCTTTTGCAAAAAGCTCCCGGGAGCTTGTATATCCATTTTCGGATCTGATCAAGAGACAGGATGAGGATCGTTTCGCATGACCGAGTACAAGCCCACGGTGCGCCTCGCCACCCGCGACGACGTCCCCAGGGCCGTACGCACCCTCGCCGCCGCGTTCGCCGACTACCCCGCCACGCGCCACACCGTCGATCCGGACCGCCACATCGAGCGGGTCACCGAGCTGCAAGAACTCTTCCTCACGCGCGTCGGGCTCGACATCGGCAAGGTGTGGGTCGCGGACGACGGCGCCGCGGTGGCGGTCTGGACCACGCCGGAGAGCGTCGAAGCGGGGGCGGTGTTCGCCGAGATCGGCCCGCGCATGGCCGAGTTGAGCGGTTCCCGGCTGGCCGCGCAGCAACAGATGGAAGGCCTCCTGGCGCCGCACCGGCCCAAGGAGCCCGCGTGGTTCCTGGCCACCGTCGGCGTCTCGCCCGACCACCAGGGCAAGGGTCTGGGCAGCGCCGTCGTGCTCCCCGGAGTGGAGGCGGCCGAGCGCGCCGGGGTGCCCGCCTTCCTGGAGACCTCCGCGCCCCGCAACCTCCCCTTCTACGAGCGGCTCGGCTTCACCGTCACCGCCGACGTCGAGGTGCCCGAAGGACCGCGCACCTGGTGCATGACCCGCAAGCCCGGTGCCTGAGCGGGACTCTGGGGTTCGAAATGACCGACCAAGCGACGCCCAACCTGCCATCACGAGATTTCGATTCCACCGCCGCCTTCTATGAAAGGTTGGGCTTCGGAATCGTTTTCCGGGACGCCGGCTGGATGATCCTCCAGCGCGGGGATCTCATGCTGGAGTTCTTCGCCCACCCCAACTTGTTTATTGCAGCTTATAATGGTTACAAATAAAGCAATAGCATCACAAATTTCACAAATAAAGCATTTTTTTCACTGCATTCTAGTTGTGGTTTGTCCAAACTCATTAATGTATCTTATCATGTCTGTATACCGTCGACCTCTAGCTGCTTGTGGAAGGCTACTCGAAATGTTTGACCCAAGTTAAACAATTTAAAGGCAATGCTACCAAATACTAATTGAGTGTATGTAAACTTCTGACCCACTGGGAATGTGATGAAAGAAATAAAAGCTGAAATGAATCATTCTCTCTACTATTATTCTGATATTTCACATTCTTAAAATAAAGTGGTGATCCTAACTGACCTAAGACAGGGAATTTTTACTAGGATTAAATGTCAGGAATTGTGAAAAAGTGAGTTTAAATGTATTTGGCTAAGGTGTATGTAAACTTCCGACTTCAACTGTATAGGGTTCCTCTA
<223> Аминокислотная последовательность рекомбинантного белка RBDomik
<210> 3
<211> 234
<400>
1 VEKGIYQTSN FRVQPTESIV RFPNITNLCP FDEVFNATRF ASVYAWNRKR
51 ISNCVADYSV LYNLAPFFTF KCYGVSPTKL NDLCFTNVYA DSFVIRGDEV
101 RQIAPGQTGN IADYNYKLPD DFTGCVIAWN SNKLDSKVSG NYNYLYRLFR
151 KSNLKPFERD ISTEIYQAGN KPCNGVAGFN CYFPLKSYSF RPTYGVGHQP
187 YRVVVLSFEL LHAPATVCGP KKSTNLVKNK CVNFNFN
Изобретение относится к биотехнологии. Описана плазмида pVBL-RBDomik, содержащая нуклеотидную последовательность, кодирующую белок RBD SARS-CoV-2 линии B.1.1.529, и обеспечивающая экспрессию белка этого RBD в клетках млекопитающих и дальнейшую очистку с помощью аффинной хроматографии. При этом получаемый белок RBD предназначен для непосредственного производства вакцинного препарата против SARS-CoV-2 линии B.1.1.529. Изобретение может быть использовано для получения белка RBD - рекомбинантного структурного домена гликопротеина spike сарбековируса SARS-CoV-2 линии B.1.1.529. 1 ил., 1 табл., 4 пр.
Рекомбинантная плазмида pVBL-RBDomik, предназначенная для синтеза и секреции белка RBD вируса SARS-CoV-2 линии B.1.617.2 в клетках млекопитающих, имеющая нуклеотидную последовательность SEQ ID NO: 2
TCAATACTGACCATTTAAATCATACCTGACCTCCATAGCAGAAAGTCAAAAGCCTCCGACCGGAGGCTTTTGACTTGATCGGCACGTAAGAGGTTCCAACTTTCACCATAATGAAATAAGATCACTACCGGGCGTATTTTTTGAGTTATCGAGATTTTCAGGAGCTAAGGAAGCTAAAATGAGCCATATTCAACGGGAAACGTCTTGCTCGAGGCCGCGATTAAATTCCAACATGGATGCTGATTTATATGGGTATAAATGGGCTCGCGATAATGTCGGGCAATCAGGTGCGACAATCTATCGATTGTATGGGAAGCCCGATGCGCCAGAGTTGTTTCTGAAACATGGCAAAGGTAGCGTTGCCAATGATGTTACAGATGAGATGGTCAGGCTAAACTGGCTGACGGAATTTATGCCTCTTCCGACCATCAAGCATTTTATCCGTACTCCTGATGATGCATGGTTACTCACCACTGCGATCCCAGGGAAAACAGCATTCCAGGTATTAGAAGAATATCCTGATTCAGGTGAAAATATTGTTGATGCGCTGGCAGTGTTCCTGCGCCGGTTGCATTCGATTCCTGTTTGTAATTGTCCTTTTAACGGCGATCGCGTATTTCGTCTGGCTCAGGCGCAATCACGAATGAATAACGGTTTGGTTGGTGCGAGTGATTTTGATGACGAGCGTAATGGCTGGCCTGTTGAACAAGTCTGGAAAGAAATGCATAAGCTTTTGCCATTCTCACCGGATTCAGTCGTCACTCATGGTGATTTCTCACTTGATAACCTTATTTTTGACGAGGGGAAATTAATAGGTTGTATTGATGTTGGACGAGTCGGAATCGCAGACCGATACCAGGATCTTGCCATCCTATGGAACTGCCTCGGTGAGTTTTCTCCTTCATTACAGAAACGGCTTTTTCAAAAATATGGTATTGATAATCCTGATATGAATAAATTGCAGTTTCACTTGATGCTCGATGAGTTTTTCTAATGAGGGCCCAAATGTAATCACCTGGCTCACCTTCGGGTGGGCCTTTCTGCGTTGCTGGCGTTTTTCCATAGGCTCCGCCCCCCTGACGAGCATCACAAAAATCGATGCTCAAGTCAGAGGTGGCGAAACCCGACAGGACTATAAAGATACCAGGCGTTTCCCCCTGGAAGCTCCCTCGTGCGCTCTCCTGTTCCGACCCTGCCGCTTACCGGATACCTGTCCGCCTTTCTCCCTTCGGGAAGCGTGGCGCTTTCTCATAGCTCACGCTGTAGGTATCTCAGTTCGGTGTAGGTCGTTCGCTCCAAGCTGGGCTGTGTGCACGAACCCCCCGTTCAGCCCGACCGCTGCGCCTTATCCGGTAACTATCGTCTTGAGTCCAACCCGGTAAGACACGACTTATCGCCACTGGCAGCAGCCACTGGTAACAGGATTAGCAGAGCGAGGTATGTAGGCGGTGCTACAGAGTTCTTGAAGTGGTGGCCTAACTACGGCTACACTAGAAGAACAGTATTTGGTATCTGCGCTCTGCTGAAGCCAGTTACCTCGGAAAAAGAGTTGGTAGCTCTTGATCCGGCAAACAAACCACCGCTGGTAGCGGTGGTTTTTTTGTTTGCAAGCAGCAGATTACGCGCAGAAAAAAAGGATCTCAAGAAGATCCTTTGATTTTCTACCGAAGAAAGGCCCACCCGTGAAGGTGAGCCAGTGAGTTGATTGCAGTCCAGTTACGCTGGAGTCTGAGGCTCGTCCTGAATGGATGGAAGTTTACATACACTTAAGTTGGAGTCATTAAAACTCGTTTTTCAACTACTCCACAAATTTCTTGTTAACAAACAATAGTTTTGGCAAGTCAGTTAGGACATCTACTTTGTGCATGACACAAGTCATTTTTCCAACAATTGTTTACAGACAGATTATTTCACTTATAATTCACTGTATCACAATTCCAGTGGGTCAGAAGTTTACATACACTAAGTTGACTGTGCCTTTAAACAGCTTGGAAAATTCCAGAAAATGATGTCATGGCTTTAGAAGCCTGCAGGCGTTACATAACTTACGGTAAATGGCCCGCCTGGCTGACCGCCCAACGACCCCCGCCCATTGACGTCAATAATGACGTATGTTCCCATAGTAACGCCAATAGGGACTTTCCATTGACGTCAATGGGTGGAGTATTTACGGTAAACTGCCCACTTGGCAGTACATCAAGTGTATCATATGCCAAGTACGCCCCCTATTGACGTCAATGACGGTAAATGGCCCGCCTGGCATTATGCCCAGTACATGACCTTATGGGACTTTCCTACTTGGCAGTACATCTACGTATTAGTCATCGCTATTACCATGATGATGCGGTTTTGGCAGTACATCAATGGGCGTGGATAGCGGTTTGACTCACGGGGATTTCCAAGTCTCCACCCCATTGACGTCAATGGGAGTTTGTTTTGACTAGTCAGGGCCCCAACCCCCCCAAGCCCCCATTTCACAACACGCTGGCGCTACAGGCGCGTGACTTCCCCTTGCTTTGGGGCGGGGGGCTGAGACTCCTATGTGCTCCGGATTGGTCAGGCACGGCCTTCGGCCCCGCCTCCTGCCACCGCAGATTGGCCGCTAGGCCTCCCCGAGCGCCCTGCCTCCGAGGGCCGGCGCACCATAAAAGAAGCCGCCCTAGCCACGTCCCCTCGCAGTTCGGCGGTCCCGCGGGTCTGTCTCAAGCTTGCCGCCAGAACACAGGTAAGTGCCGTGTGTGGTTCCCGCGGGCCTGGCCTCTTTACGGGTTATGGCCCTTGCGTGCCTTGAATTACTTCCATGCCCCTGGCTGCAGTACGTGATTCTTGATCCCGAGCTTCGGGTTGGAAGTGGGTGGGAGAGTTCGAGGCCTTGCGCTTAAGGAGCCCCTTCGCCTCGTGCTTGAGTTGAGGCCTGGCTTGGGCGCTGGGGCCGCCGCGTGCTAATCTGGTGGCACCTTCGCGCCTGTCTCGCTGCTTTCGCTAAGTCTCTAGCCATTTAAAATTTTTGATAACCAGCTGCGACGCTTTTTTTCTGGCGAGATAGTCTTGTAAATGCGGGCCAAGATCTGCACACTGGTATTTCGGTTTTTGGGGCCGCGGGCGGCGACGGGGCCCGTGCGTCCCAGCGCACATGTTCGGCGAGGCGGGGCCTGCGAGCGCGGCCACCGAGAATCGGACGGGGGTAGTCTCAAACTGGCCGGCCTGCTCTGGTGCCTGGCCTCGCGCCGCCGTGTATCGCCCCGCCCTGGGCGGCAAGGCTGGCCCGGTCGGCACCAGTTGCGTGAGCGGAAAGATGGCCGCTTCCCGGCCCTGCTGCAGGGAGCTCAAAATGGAGGACGCGGCGCCCGGGAGAGCGGGCGGGTGAGTCACCCACACAAAGGAAAAGGGCCTTTCCTTCCTCATCCGTCGCTTCATGTGACTCCACGGAGTACCGGGCGCCGTCCAGGCACCTCGATTAGTTCTCGAGCTTTTGGAGTACGTCGTCTTTAGGTTGGGGGGAGGGGTTTTATGCGATGGAGTTTCCCCACACTGAGTGGGTGGAGACTGAAGAGTTAGGCCAGCTTGGCACTTGATGTAATTCTCCTTGGAATTTGCCCTTTTTGAGTTTGGATCTTGCCTCATTCTCAAGCCTCAGACAGTGGTTCAAAGTTTTTTTCTTCCATTTCAGCTCCTGGGCAACGTTAGCCTCGAGAATTCGTCCTGCTGCGCACGTGGGAAGCCCTGGCCCCGGCCGCCACCATGGGAGTGAAGGTGCTGTTCGCCCTGATCTGTATTGCCGTGGCCGAAGCTTCTGTGGAAAAGGGCATCTACCAGACCAGCAACTTCCGGGTGCAGCCCACCGAATCCATCGTGCGGTTCCCCAATATCACCAATCTGTGCCCCTTCGACGAGGTGTTCAATGCCACCAGATTCGCCTCTGTGTACGCCTGGAACCGGAAGCGGATCAGCAATTGCGTGGCCGACTACTCCGTGCTGTACAACCTGGCCCCCTTCTTCACCTTCAAGTGCTACGGCGTGTCCCCTACCAAGCTGAACGACCTGTGCTTCACAAACGTGTACGCCGACAGCTTCGTGATCCGGGGAGATGAAGTGCGGCAGATTGCCCCTGGACAGACAGGCAACATCGCCGACTACAACTACAAGCTGCCCGACGACTTCACCGGCTGTGTGATTGCCTGGAACAGCAACAAGCTGGACTCCAAAGTCTCCGGCAACTACAATTACCTGTACCGGCTGTTCCGGAAGTCCAATCTGAAGCCCTTCGAGCGGGACATCTCCACCGAGATCTATCAGGCCGGCAACAAGCCTTGTAACGGCGTGGCCGGCTTCAACTGCTACTTCCCACTGAAGTCCTACTCCTTTCGGCCCACATACGGCGTGGGCCACCAGCCCTACAGAGTGGTGGTGCTGAGCTTCGAACTGCTGCATGCCCCTGCCACAGTGTGCGGCCCTAAGAAAAGCACCAATCTCGTGAAGAACAAATGCGTGAACTTCAACTTCAACACCTACATTCCTGAGGCTCCCAGAGATGGCAAGGCCTACGTCAGAAAGGATGGCGCTTGGGTCGACCTGGAAACCCTGTTGCCTTAAGTCGACCGAGCTCAGCCTCGACTGTGCCTTCTAGTTGCCAGCCATCTGTTGTTTGCCCCTCCCCCGTGCCTTCCTTGACCCTGGAAGGTGCCACTCCCACTGTCCTTTCCTAATAAAATGAGGAAATTGCATCGCATTGTCTGAGTAGGTGTCATTCTATTCTGGGGGGTGGGGTGGGGCAGGACAGCAAGGGGGAGGATTGGGAAGACAATAGCAGGCATGCTGGGGATGCGGTGGGCTCTATGGCTTCTGAGGCGGAAAGAACCAGCTGGGGCTCTAGGGGGTATCCCCACGCGCCCTGTAGCGGCGCATTAAGCGCGGCGGGTGTGGTGGTTACGCGCAGCGTGACCGCTACACTTGCCAGCGCCCTAGCGCCCGCTCCTTTCGCTTTCTTCCCTTCCTTTCTCGCCACGTTCGCCGGCTTTCCCCGTCAAGCTCTAAATCGGGGGCTCCCTTTAGGGTTCCGATTTAGTGCTTTACGGCACCTCGACCCCAAAAAACTTGATTAGGGTGATGGTTCACGTAGTGGGCCATCGCCCTGATAGACGGTTTTTCGCCCTTTGACGTTGGAGTCCACGTTCTTTAATAGTGGACTCTTGTTCCAAACTGGAACAACACTCAACCCTATCTCGGTCTATTCTTTTGATTTATAAGGGATTTTGCCGATTTCGGCCTATTGGTTAAAAAATGAGCTGATTTAACAAAAATTTAACGCGAATTAATTCTGTGGAATGTGTGTCAGTTAGGGTGTGGAAAGTCCCCAGGCTCCCCAGCAGGCAGAAGTATGCAAAGCATGCATCTCAATTAGTCAGCAACCAGGTGTGGAAAGTCCCCAGGCTCCCCAGCAGGCAGAAGTATGCAAAGCATGCATCTCAATTAGTCAGCAACCATAGTCCCGCCCCTAACTCCGCCCATCCCGCCCCTAACTCCGCCCAGTTCCGCCCATTCTCCGCCCCATGGCTGACTAATTTTTTTTATTTATGCAGAGGCCGAGGCCGCCTCTGCCTCTGAGCTATTCCAGAAGTAGTGAGGAGGCTTTTTTGGAGGCCTAGGCTTTTGCAAAAAGCTCCCGGGAGCTTGTATATCCATTTTCGGATCTGATCAAGAGACAGGATGAGGATCGTTTCGCATGACCGAGTACAAGCCCACGGTGCGCCTCGCCACCCGCGACGACGTCCCCAGGGCCGTACGCACCCTCGCCGCCGCGTTCGCCGACTACCCCGCCACGCGCCACACCGTCGATCCGGACCGCCACATCGAGCGGGTCACCGAGCTGCAAGAACTCTTCCTCACGCGCGTCGGGCTCGACATCGGCAAGGTGTGGGTCGCGGACGACGGCGCCGCGGTGGCGGTCTGGACCACGCCGGAGAGCGTCGAAGCGGGGGCGGTGTTCGCCGAGATCGGCCCGCGCATGGCCGAGTTGAGCGGTTCCCGGCTGGCCGCGCAGCAACAGATGGAAGGCCTCCTGGCGCCGCACCGGCCCAAGGAGCCCGCGTGGTTCCTGGCCACCGTCGGCGTCTCGCCCGACCACCAGGGCAAGGGTCTGGGCAGCGCCGTCGTGCTCCCCGGAGTGGAGGCGGCCGAGCGCGCCGGGGTGCCCGCCTTCCTGGAGACCTCCGCGCCCCGCAACCTCCCCTTCTACGAGCGGCTCGGCTTCACCGTCACCGCCGACGTCGAGGTGCCCGAAGGACCGCGCACCTGGTGCATGACCCGCAAGCCCGGTGCCTGAGCGGGACTCTGGGGTTCGAAATGACCGACCAAGCGACGCCCAACCTGCCATCACGAGATTTCGATTCCACCGCCGCCTTCTATGAAAGGTTGGGCTTCGGAATCGTTTTCCGGGACGCCGGCTGGATGATCCTCCAGCGCGGGGATCTCATGCTGGAGTTCTTCGCCCACCCCAACTTGTTTATTGCAGCTTATAATGGTTACAAATAAAGCAATAGCATCACAAATTTCACAAATAAAGCATTTTTTTCACTGCATTCTAGTTGTGGTTTGTCCAAACTCATTAATGTATCTTATCATGTCTGTATACCGTCGACCTCTAGCTGCTTGTGGAAGGCTACTCGAAATGTTTGACCCAAGTTAAACAATTTAAAGGCAATGCTACCAAATACTAATTGAGTGTATGTAAACTTCTGACCCACTGGGAATGTGATGAAAGAAATAAAAGCTGAAATGAATCATTCTCTCTACTATTATTCTGATATTTCACATTCTTAAAATAAAGTGGTGATCCTAACTGACCTAAGACAGGGAATTTTTACTAGGATTAAATGTCAGGAATTGTGAAAAAGTGAGTTTAAATGTATTTGGCTAAGGTGTATGTAAACTTCCGACTTCAACTGTATAGGGTTCCTCTA
Авторы
Даты
2022-05-26—Публикация
2021-12-13—Подача