Изобретение относится к интегративному плазмидному вектору pVEAL3-RBDdel, обеспечивающему синтез и секрецию рекомбинантного белка рецепторсвязывающего домена RBDdelta коронавируса SARS-CoV-2 в клетках млекопитающих, рекомбинантному штамму клеточной линии СНО-К1-RBDdelta и рекомбинантному белку RBDdelta SARS-CoV-2, продуцируемому указанным штаммом клеточной линии и может быть использовано в генной инженерии, биотехнологии и медицине. Рекомбинантный белок RBDdelta может служить, в первую очередь, для создания вакцины против новой коронавирусной инфекции COVID-19, а также для создания диагностикумов.
Уровень техники.
В последнее десятилетие для создания вакцины против возбудителя атипичной пневмонии SARS-CoV, был реализован ряд подходов, включая использование инактивированного цельного вируса, рекомбинантного белка S SARS-CoV и рецепторсвязывающий домен (RBD) белка S. Был разработан ряд вакцин, содержащие инактивированный цельный вирус, в комплексе с адъювантом Alhydrogel©, однако, у мышей иммунизированных этой конструкцией наблюдалась эозинофильная патология, вероятно, из-за антител озависимого усиления инфекции (ADE) [1,2]. Позднее были разработаны вакцины с использованием рекомбинантного S-белка SARS-CoV, но полноразмерный S-белок, по-видимому, так же индуцирует ADE [3]. В качестве альтернативного подхода, был апробирован подход на основе RBD белка S. В молекуле поверхностного гликопротеина SARS-CoV и MERS, выделяют два основных региона уязвимости RBD и HR1 (регион пептида слияния) [4-8]. Рекомбинантный RBD, в комплексе с адъювантом Фрейнда и с системой адъювантов Sigma вызывает индукцию нейтрализующих антител и формирование протективного иммунитета у вакцинированных животных, без ADE и других побочных иммунных реакций [6,9].
В настоящее время накопилось достаточно свидетельств того, что вакцина на основе RBD показывает высокий уровень иммуногенности, при низкой реактогенной и хорошей переносимости. В первую очередь это следует из результатов клинических испытаний. По данным на 30 марта 2023 года, 57 субъединичных вакцин находятся на стадии клинических испытаний. Из них 23 проходят III фазу клинических испытаний, [https://www.who.int/publications/ m/item/draft-landscape-of-covid-19-candidate-vaccines]. Отчет об испытаниях вакцины на основе RBD, свидетельствует об отсутствии серьезных побочных эффектов при использовании трехкратной схемы введения (0, 30 и 60 сутки), в то время как уровень сероконверсии составляет 93-100% [NCT04466085]. Еще 20 вариантов субъединичных вакцин проходят I и II фазы клинических испытаний [https://www.who.int/publications/ m/item/draft-landscape-of-covid-19-candidate-vaccines].
Использование только последовательности RBD в качестве вакцины позволяет добиться появления высоких титров нейтрализующих антител, этот факт тем более примечателен что использование тримера S или фрагмента S1 дает сходные или худшие результаты [10-12].
Ближайшие аналоги.
Известна заявка на изобретение Китая (CN 111217917, МПК А61К 39/215, опубл. 02.06.2020 г.), в которой описан способ получения слитых белков RBD-CTB и RBDCRM 197(A) для создания рекомбинантных субъединичных вакцин для лечения и профилактики COVID-19. Для этого в экспрессионный вектор рЕТ9а встраивали нуклеотидные последовательности, кодирующие RBD соединенные линкером с адъювантами СТВ или CRM 197(A), и проводили трансформацию клеток Escherichia coli BL21 полученными конструкциями. Недостатком данного изобретения является выбор системы экспрессии в клетках Escherichia coli BL21, поскольку известно, что у прокариот отсутствуют некоторые системы посттрансляционной модификации белков, например, гликозилирование, необходимое для получения корректной формы RBD, так же существует риск некорректного фолдинга молекул. Такие особенности могут критически снижать уровень иммуногенности итоговой вакцины.
Известна заявка на изобретение Китая (CN 111254155, МПК A61K 39/215, опубл. 09.06.2020 г. ), в которой варианты RBD для субъединичных вакцин против вируса SARS-CoV-2 получали с использованием растений-хозяев. Авторы с помощью транзиентной экспресии в Lactuca sativa (салат-латук) получали слитые белки CTB-RBD и RBD-Fc, используемые в вакцине для профилактики COVID-19. Недостатком изобретения является способ выделения целевых белков, включающий многоступенчатую систему очистки, а также особенности экспрессии, связанные с некорректным гликозилированием и фолдингом белков в растительных системах экспрессии.
Известен аналог (патент РФ №2723008, МПК C07K 14/165, C12N 15/50, опубл. 08.06.2020 г. ), содержащий:
- генетическую конструкцию для экспрессии рекомбинантного белка RBD вируса SARS-CoV-2, включающую последовательность SEQ ID NO: 1;
- штамм клеток яичника китайского хомячка CHO-S-RBD - продуцент рекомбинантного белка RBD вируса SARS-CoV-2, содержащий генетическую конструкцию, включающую SEQ ID NO: 1;
- способ получения штамма клеток яичника китайского хомячка CHO-S-RBD, включающий: получение генетической конструкции, включающей SEQ ID NO: 1; введение указанной генетической конструкции в клетки путем липофекции; селекцию клеток на антибиотике Hygromycin В;
- рекомбинантный белок RBD вируса SARS-CoV-2 для выявления антител к SARSCoV-2, имеющий последовательность SEQ ID NO: 2, являющийся продуктом штамма CHO-S-RBD;
- способ получения рекомбинантного белка RBD вируса SARS-CoV-2, включающий: культивирование штамма клеток яичника китайского хомячка CHO-S-RBD; хроматографическую очистку рекомбинантного белка RBD вируса SARS-CoV-2 из культуральной среды штамма клеток яичника китайского хомячка CHO-S-RBD; подтверждение получения рекомбинантного белка RBD вируса SARS-CoV-2, имеющего SEQ ID NO: 2.
Недостатком изобретения является использование относительно слабого лидерного пептида тяжелой цепи иммуноглобулина G человека, что может снизить выход целевого белка. Кроме того, негативное влияние на выход продукта может оказать тот факт, что не проведена оптимизация кодонного состава нуклеотидной последовательности, кодирующей RBD, для продуцента, а также использован вектор для эписомальной (внехромомсомной) экспрессии. Авторами показан довольно низкий уровень продукции 5-10 мг/л суспензионной культуры.
Известна рекомбинантная плазмида pVNV-GL-RBDind, обеспечивающая синтез и секрецию рекомбинантного рецепторсвязывающего домена (RBD) коронавируса SARS-CoV-2 линии В. 1.617 в клетках млекопитающих (патент РФ №2754930, МПК C12N 15/50, опубл. 09.09.2021 г.).
К недостаткам данной генетической конструкции следует отнести используемый дизайн ДНК, кодирующей последовательность RBD, не учитывающий современные представления о молекулярно-генетическом полиморфизме вируса SARS-CoV-2 вследствие чего концентрация белка RBD составляет 5-10 мг на литр культуральной среды.
Наиболее близким аналогом (прототипом) является изобретение (патент РФ №2752858, МПК C12N 15/74, опубл. 11.08.2021 г.), которое содержит:
- интегративный плазмидный вектор pVEAL2-S-RBD, обеспечивающий экспрессию и секрецию белка RBD коронавируса SARS-CoV-2 в клетках млекопитающих;
- рекомбинантный штамм клеточной линии яичника китайского хомячка CHO-K1-RBD продуцента рекомбинантного белка RBD вируса SARS-CoV-2, содержащий интегративный плазмидный вектор pVEAL2-S-RBD, имеющий нуклеотидную последовательность SEQ ID NO:2, обеспечивающий экспрессию и секрецию белка RBD коронавируса SARS-CoV-2;
- рекомбинантный белок RBD коронавируса SARS-CoV-2, продуцируемый рекомбинантным штаммом клеточной линии яичника китайского хомячка CHO-K1-RBD, имеющий аминокислотную последовательность SEQ ID NO:3 и предназначенный для создания иммунобиологических препаратов.
К недостаткам данной генетической конструкции следует также отнести используемый дизайн ДНК, кодирующей последовательность RBD, не в полной мере учитывающий современные представления о молекулярно-генетическом полиморфизме вируса SARS-CoV-2 вследствие чего концентрация белка RBD варианта Wuhan SARS-CoV-2 составляла 50-100 мг с литра культуральной среды.
Техническим результатом заявленного изобретения является повышение выхода целевого рекомбинантного белка RBDdelta SARS-CoV-2 за счет:
- создания кодон-оптимизированной нуклеотидной последовательности, кодирующей белок RBDdelta SARSCoV-2 для экспрессии в клетках СНО, конструирования интегративного плазмидного вектора pVEAL3-RBDdelta, содержащего кодон-оптимизированный ген RBDdelta и обеспечивающего его экспрессию в клетках млекопитающих, и разработки с помощью указанного вектора штамма-продуцента рекомбинантного белка RBDdelta SARS-CoV-2 на основе рекомбинантного штамма клеточной линии яичников китайского хомячка СНО-К1;
адаптации клона №33 культуры-продуцента СНО-K1-RBDdelta, показавший наибольшую продуктивность при адгезионном культивировании к суспензионному культивированию.
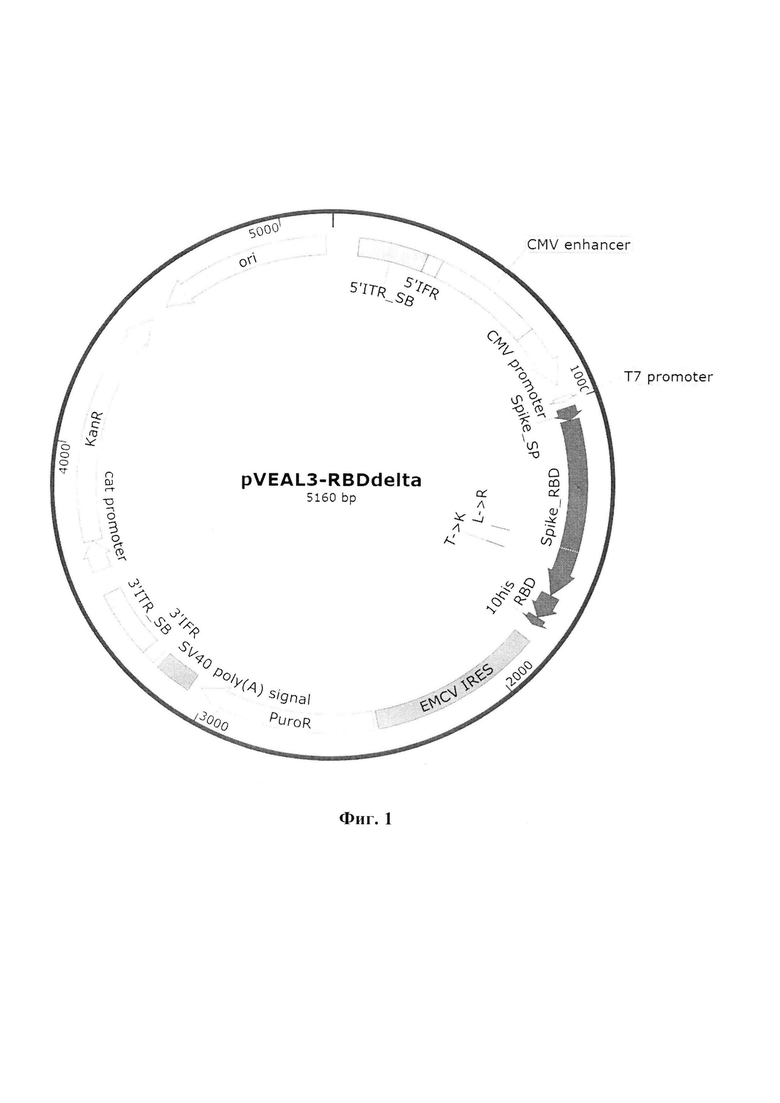
Технический результат достигается созданием интегративного плазмидного вектора pVEAL3-RBDdelta, обеспечивающего синтез и секрецию белка RBDdelta коронавируса SARS-CoV-2 в клетках млекопитающих, имеющего размер 5160 п. н., нуклеотидную последовательность SEQ ID NO:l (фиг. 5) и содержащего в соответствии с физической и генетической картой, представленной на фиг. 1, следующие конструктивные элементы:
- участок начала репликации ori, имеющий координаты с 4552 по 5139;
- последовательности прямых и инвертированных повторов IR/DR, содержащие сайты связывания с транспозазой SB 100, имеющие координаты с 88 по 368 и с 3210 по 3487;
- CMV энхансер, имеющий координаты с 369 по 733 и CMV промотор, имеющий координаты с 734 по 937 и являющийся наиболее часто используемым промотором в генно-терапевтических конструкциях;
- лидерный пептид S-SP, обеспечивающий экспорт белка из клеток СНО-K1- RBDdelta и имеющий координаты с 3090 по 3134;
- кодон-оптимизированная нуклеотидная последовательность SEQ ID N0:2 (фиг. 6) гена, кодирующего рецепторсвязывающий домен RBDdelta коронавируса SARS-CoV-2, в которую внесены 2 мутации для получения белка RBDdelta варианта В. 1.617.2 и которая имеет координаты с 1066 по 1767;
- последовательность 10xHis, имеющая координаты с 1768 по 1797 и являющаяся полигистидиновым тэгом для очистки рекомбинантного белка с помощью металл-хеллатной хроматографии;
- участок внутренней посадки рибосомы EMCV IRES, имеющий координаты с 1847 по 2421;
- последовательность PuroR, кодирующая фактор устойчивости к антибиотику пуромицину, имеющая координаты с 2434 по 3033;
- последовательность SV40 poly(A) signal для стабилизации мРНК-транскриптов за счет полиаденилирования, имеющая координаты с 3068 по 3189;
- промотор cat синтетический бактериальный промотор, имеющий координаты с 3571 по 3673;
- нуклеотидная последовательность KanR, кодирующая фактор устойчивости к антибиотику канамицину, позволяющая проводить амплификацию плазмиды в E.coli и имеющая координаты с 3674 по 4489.
Указанный технический результат достигается также созданием рекомбинантного штамма клеточной линии яичника китайского хомячка СНО-Kl-RBDdelta продуцента рекомбинантного белка рецепторсвязывающего домена RBDdelta коронавируса SARS-CoV-2, содержащего интегративный плазмидный вектор pVEAL3-RBDdelta по п. 1, и депонированного под номером 327 в коллекции культур клеток ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора.
Указанный технический результат достигается также получением рекомбинантного белка RBDdelta коронавируса SARS-CoV-2, продуцируемого рекомбинантным штаммом клеточной линии яичника китайского хомячка СНО-К1-RBDdelta по п. 2, имеющего аминокислотную последовательность SEQ ID NO: 3 (фиг. 7) и предназначенного для создания иммунобиологических препаратов.
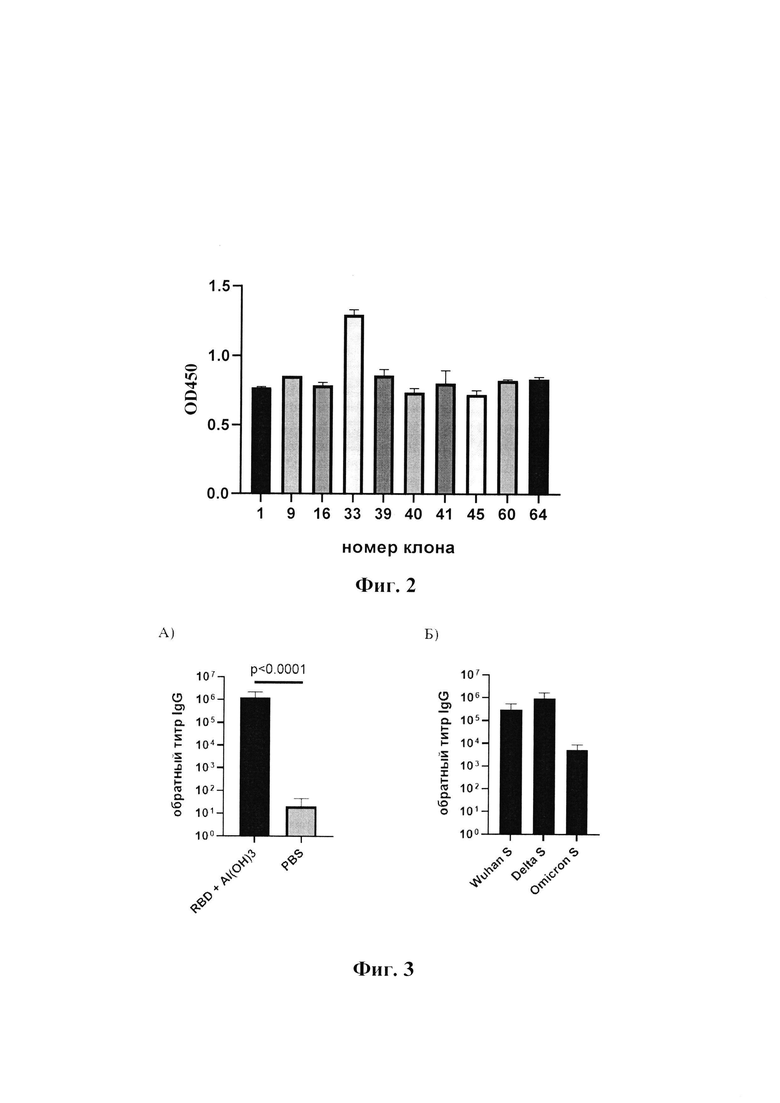
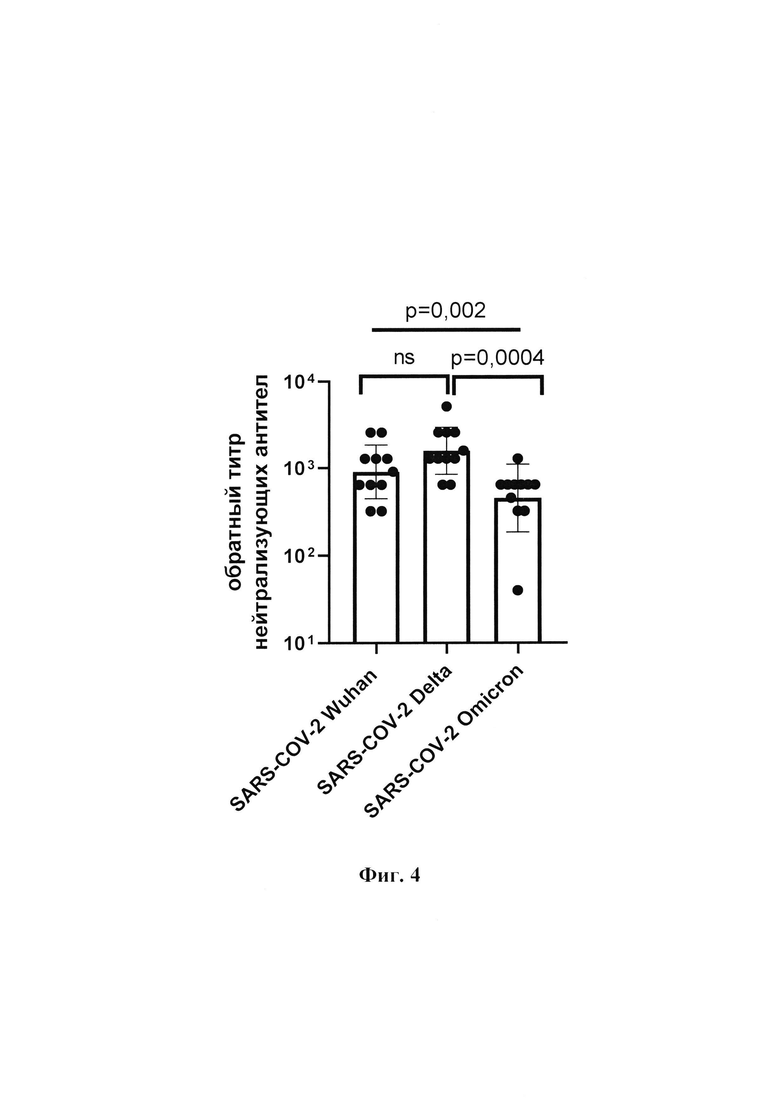
Изобретение поясняется графическими материалами, представленными на фиг.1,2,3,4 и таблицами 1. На фиг. 1 изображена физическая и генетическая карта универсального рекомбинантного вектора pVEAL3-RBDdelta. На фиг. 2 приведена относительная продуктивность клонов СНО-К1-RBDdelta в ИФА. На фиг. 3. представлены данные анализа сывороточных IgG у мышей иммунизированных 50 мкг RBD Delta SARS-CoV-2 с гидроокисью алюминия по результатам ИФА. На фиг. 4 представлены данные анализа сывороточных IgG у мышей иммунизированных дозой 50 мкг RBD Delta SARS-CoV-2 с гидроокисью алюминия в тесте на ингибирование цитопатического действия вариантов Delta(B. 1.617.2), Wuhan и Omicron (В. 1.1.529) вируса SARS-CoV-2 (100 ТЦД50) на культуре клеток Vero Е6. На фиг. 5 представлена нуклеотидная последовательность интеграционного вектора pVEAL3-RBDdelta. На фиг.6 приведена нуклеотидная последовательность гена, кодирующая рекомбинантный рецепторсвязывающего домен (RBDdelta) коронавируса SARS-CoV-2. На фиг. 7 представлена аминокислотная последовательность белка рекомбинантного рецепторсвязывающего домена (RBDdelta) коронавируса SARS-CoV-2.
Для лучшего понимания сущности предлагаемого изобретения ниже приведены примеры его осуществления. Все стандартные генно-инженерные и микробиологические манипуляции, а также амплификацию и секвенирование ДНК проводили по известным методикам [13].
Пример 1. Конструирование интегративного плазмидного вектора pVEAL3-RBDdelta для синтеза и секреции белка RBD delta SARS-CoV-2 в клетках млекопитающих
Ранее в нашей лаборатории на основе клеток СНО-К1 был разработан штамм CHO-K1-RBD, продуцент RBD варианта Wuhan SARS-CoV-2 (патент РФ №2752858).
Для получения RBD варианта Delta SARS-CoV-2 в нуклеотидную последовательность, кодирующую RBD в векторе pVEAL2-RBD, вводили нуклеотидные замены для получения аминокислотных замен (L452R и Т479К).
На матрице RBD Wuhan проводили ПЦР с использованием праймеров T479K F и URBD-R, а также URBD-F и T479K_R, а затем отжиг продуктов реакции и ПЦР с праймерами URBD-F и URBD-R (таблица 1). Итоговый продукт гидролизовали рестриктазами Bspl3I и AspA2I. В таблице 1 представлены последовательности синтезированных олигонуклеотидов, использованных для получения плазмиды pVEAL3-RBDdelta.
Отдельно матрице RBD Wuhan проводили ПЦР с использованием праймеров URBD-F и L452R-R и гидролиз рестрикатазами AsuNHI and Bspl3I.
Затем вектор pVEAL2-RBD гидролизовали по сайтам AsuNHI и AspA2I и проводили одновременную встройку двух фрагментов. Подтверждение структуры плазмиды проводили секвенированием по методу Сенгера. Затем, чтобы уменьшить размер плазмиды из вектора удаляли элементы UCOE. Вектор получил название pVEAL3-RBDdel (на фиг. 1 представлена физическая и генетическая карта интегративного плазмидного вектора pVEAL3-RBDdelta). Нуклеотидная последовательность SEQ ID NO:l вектора pVEAL3-RBDdel приведена на фиг.5 и в приложении.

Пример 2. Получение штамма рекомбинантной клеточной линии CHO-Kl-RBDdelta, продуцента рекомбинантного домена RBD delta SARS-CoV-2.
Рекомбинантную клеточную линию CHO-Kl-RBDdelta, продуцент рекомбинантного домена RBD Delta SARS-CoV-2, получали на основе клеточной линии яичников китайского хомячка СНО-К1 с использованием разработанной конструкции pVEAL3-RBDdelta. Клетки растили в инкубаторе при 5% содержания С02, 80%-ной влажности. При достижении 80% плотности монослоя проводили трансфекцию клеток плазмидой pVEAL3-RBDdelta с помощью Lipofectamine 3000 (ThermoFisher, США) в соответствии с инструкцией производителя. Для интеграции экспрессионной кассеты вектора pVEAL3-RBDdelta в геном клеток совместно с плазмидой pVEAL3-RBDdelta добавляли плазмиду pCMV(CAT)T7-SB100, кодирующую транспозазу SB 100, в отношении 10:1. Через 3 дня в культуральную среду добавляли селективный антибиотик пуромицин (InvivoGen, США) в конечной концентрации 10 мкг/мл, ген устойчивости к которому входит в состав вектора.
Селекцию устойчивых клонов проводили в течение трех суток, затем поликлональную клеточную культуру рассевали в 96 луночном планшете в концентрации 1 клетка на лунку. Спустя две недели, анализировали наличие единичных колоний в лунках, отбирали культуральную жидкость и оценивали продуктивность клонов с помощью ИФА.
На фиг. 2 представлена относительная продуктивность клонов CHO-Kl-RBDdelta в ИФА. Клон №33 культуры-продуцента CHO-Kl-RBDdelta, показавший наибольшую продуктивность при адгезионном культивировании адаптировали к суспензионному культивированию.
Штамм депонирован 08.11.2022 г. в коллекции ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора под номером 327. Характеристика рекомбинантного штамма CHO-Kl-9E2ch (справка о депонировании прилагается).
Характеристика штамма.
Морфология: веретеновидные и эпителиоподобные клетки с круглыми ядрами, содержащими от 1 до 2 ядрышек.
Способ культивирования: монослойный.
Среда для культивирования: питательная среда DMEM/F-12 (1:1) - 90%, сыворотка крови плодов коровы - 10%.
Температура культивирования: 37°С.
Посевная концентрация: 100 тыс.клеток в 1 мл.
Метод снятия: 0,25% трипсин (1/3) и 0,02% версен (2/3).
Кратность рассева: 1:3.
Частота пассирования: 3-4 суток.
Условия криоконсервации: питательная среда DMEM/F-12 (1:1) - 50%, сыворотка крови плодов коровы - 40%, ДМСО - 10%.
Режим замораживания: при температуре 4°С выдерживают 1 ч; минус 80°С -12 ч; минус 196°С - хранение.
Условия хранения: в криопробирках в количестве 5 шт. хранится в жидком азоте при температуре минус 196°С.
Номер пассажа в жидком азоте: 3.
Жизнеспособность после криоконсервации: 85-90%.
Маркерый признак: наличие в культуральной среде рекомбинантного рецепторсвязывающего домена (RBD) S-белка SARS-CoV-2 размером ~35 кДа, подтверждается с помощью белкового электрофореза и иммуноблотинга. Область применения: биотехнология.
Пример 3. Получение рекомбинантного белка RBD delta с использованием штамма рекомбинантной клеточной линии CHO-Kl-RBDdelta. Клон №33 культуры-продуцента CHO-Kl-RBDdelta, показавший наибольшую продуктивность при адгезионном культивировании, адаптировали к суспензионному культивированию.
Затем проводили наработку препарата RBDdelta в суспензии. Клон-продуцент RBDdelta рассевали в 5 мл свежей среды HyCell СНО + 4 mM GlutaMAX с концентрацией 1×106 живых кл/мл в 50 мл центрифужные пробирки. Культивировали на шейкере 200 об/мин, орбита 19 мм, при +37°С во влажной атмосфере 5% СО2 6-7 дней до достижения концентрации 8×106 живых кл./мл. Суспензию клеток 5 мл с концентрацией 8×106 живых кл./мл инокулировали в стеклянной колбе с 35 мл свежей среды HyCell СНО + 4 mM GlutaMAX. Культивировали на шейкере при тех же условиях 6-7 дней до достижения концентрации 8×106 живых кл./мл. Суспензию клеток 5 мл с концентрацией 8×106 живых кл./мл инокулировали в новую стеклянную колбу с 35 мл свежей среды HyCell СНО + 4 mM GlutaMAX. Оставшийся объем переносили на шейкере (200 об./мин, орбита 19 м) при +31°С инкубировали 10 дней. Культуральную жидкость очищали от клеток при помощи центрифугирования 12000g при 4°С.Для получения рекомбинантного белка RBDdelta была проведена двух-стадийная хроматографическая очистка, включающая аффинную и ионообменную хроматографии. Аминокислотная последовательность SEQ ID NO:3 приведена на фиг. 7 и в приложении.
Пример 4. Оценка иммуногенности рекомбинантного белка RBD SARS-CoV-2
Оценку иммуногенности RBD проводили в экспериментах по иммунизации мышей линии BALB/c. В качестве адъюванта использовали гидроокись алюминия (А1(ОН)3).
Мышей иммунизировали внутримышечно, дважды с двухнедельным интервалом дозой 50 мкг белка RBD с гидроокисью алюминия. Через две недели после второй иммунизации получали сыворотки крови и анализировали титры специфических IgG в ИФА с использованием RBD Delta, а также тримеров S-белка вариантов Delta, Wuhan и Omicron SARS-CoV-2 (фиг. 3).
На фиг. 3 представлены данные анализа сывороточных IgG у мышей иммунизированных 50 мкг RBD Delta SARS-CoV-2 с гидроокисью алюминия по результатам ИФА.
А. Титры RBD Delta специфических IgG.
Б. Титры IgG специфических к тримерам S-белка штаммов Wuhan, Delta, Omicron.
Статистический анализ выполнен в GraphPad Prism 8.0 с использованием непараметрического теста Манна-Уитни.
По результатам анализов, показано, что иммуноген достоверно вызывает индукцию специфических IgG у мышей линии BALB/c. Кроме того, сывороточные антитела иммунизированных мышей, способны связываться с тримерами S-белка штаммов Delta и Omicron. Среднегеометрический титр (GMT) составил 2,4×105±2,1 для Wuhan S, 7,1×105±±2,5 для Delta S и 4,1×103±2,1 Omicron S.
Анализ нейтрализующей активности сывороток мышей определяли в реакции нейтрализации цитопатического действия (ЦПД) вируса на культуре клеток. В работе использовали штаммы вируса Wuhan- hCoV-19/Australia/VICO 1/2020 (EPIJSL 406844), Delta- hCoV-19/Russia/PSK-2804/2021 (EPIJSL 7338814) и Omicron 1- hCoV-19/Russia/Moscowl71619-031221/2021 (EPI_ISL_8920444) SARS-CoV-2 были получены из государственной коллекции возбудителей вирусных инфекций и риккетсиозов ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора. Вируссодержащий материал хранился при температуре -70°С.Концентрация вируса в наработанном препарате составляла 5×106 ТЦД50/мл (тканевая цитопатическая доза на миллилитр).
Образцы крови получали от всех животных через две недели после 2-й иммунизации. Из крови животных были получены сыворотки путем осаждения форменных элементов с помощью центрифугирования при 1000 × g в течение 7 - 10 мин. Полученные сыворотки термически инактивировали при 56°С в течение 30 мин.
Определение нейтрализующих титров сывороток, проводили путем учета неповрежденного монослоя культуры клеток Vero Е6 в лунках 96-луночного планшета (Corning, США). Культуру клеток выращивали в 96-луночных культуральных планшетах в ростовой питательной среде с добавлением 10% бычьей фетальной сыворотки и антибиотиков. Заражение клеток проводили вариантами Delta (В. 1.617.2), Wuhan и Omicron (В. 1.1.529) вируса SARS-CoV-2 в дозе 100 ТЦД50/лунку. Исследуемые образцы сыворотки крови разводили последовательно в среде DMEM (ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора, Россия) с глутамином и антибиотиками с 2-кратным шагом, начиная с разведения 1:10 до 1:2560. К разведениям сыворотки добавляли вирус в равной пропорции 1:1 и инкубировали в течение 1 ч при 37°С, затем смесь вируса и сыворотки наносили в дублях на монослой культуры клеток в объеме 100 мкл/лунку. Планшеты инкубировали в течение 4 суток при 37°С во влажной атмосфере 5% СО2. Для окрашивания в каждую лунку планшета вносили 100 мкл 0,2%-го раствора генцианвиолета. Через 4 сутки жидкость из лунок удаляли и промывали лунки водой. Учет результатов проводили визуально.
Любое специфическое поражение культуры клеток в лунке учитывали как цитопатическое действие (ЦПД). Титром считали последнее разведение, при котором регистрировали защиту монослоя культуры клеток в лунках от ЦПД вируса. В качестве положительного контроля использовали 20-кратное разведение образца сыворотки крови реконвалесцента COVID-19 с ранее установленным титром 1:80. В качестве отрицательного контроля использовали питательную среду.
Расчет титра нейтрализующих антител проводили по формуле Рида-Менча. Результаты эксперимента (фиг. 4) подтверждают, что заявленный рекомбинантного RBDdelta обеспечивает гуморальный ответ в отношении в SARS-CoV-2 вариантов Delta (В. 1.617.2), Wuhan, Omicron (В. 1.1.529). На фиг. 4 представлены результаты исследования Анализ сывороточных IgG у мышей иммунизированных дозой 50 мкг RBD Delta SARS-CoV-2 с гидроокисью алюминия в тесте на ингибирование цитопатического действия вариантов Delta(B. 1.617.2), Wuhan и Omicron (В. 1.1.529) вируса SARS-CoV-2 (100 ТЦД50) на культуре клеток Vero Е6.
На фиг.4 представлены данные анализа сывороточных IgG у мышей иммунизированных дозой 50 мкг RBD Delta SARS-CoV-2 с гидроокисью алюминия в тесте на ингибирование цитопатического действия вариантов Delta(B. 1.617.2), Wuhan и Omicron (В. 1.1.529) вируса SARS-CoV-2 (100 ТЦД50) на культуре клеток Vero Е6. Представлены средние геометрические значения ± SD. Статистический анализ выполнен с использованием U критерия Манна-Уитни и критерия Краскела-Уоллиса.
Таким образом, группа заявленных изобретений обеспечивает получение иммунологически корректной формы белка RBD delta SARS-CoV-2, который может быть использован как платформа для создания вакцины против COVID-19. В примерах 1-4 подтверждается достижение технического результата заявляемого изобретения, а именно: выход рекомбинантного белка RBD delta SARS-CoV-2 после хроматографической очистки составляет 150 мг с литра культуральной среды, что превосходит в 15 раз выход белка RBD, представленный в аналоге по патенту РФ №2754930 (продукция белка RBD SARS-CoV-2 в аналоге составляет от 5 мг/л до 10 мг/л). В прототипе по патенту РФ №2752858 выход рекомбинантного белка RBD SARS-CoV-2 после хроматографической очистки составляет 50-100 мг с литра культуральной среды, что в 1,5-3 раза ниже, чем в предлагаемом изобретении. Источники научно-технической информации:
1. Perlman S., Dandekar A.A. Immunopathogenesis of coronavirus infections: Implications for SARS//Nat. Rev. Immunol. - 2005. - V. 5 - №12. - P. 917-27.
2. Bolles M, Deming D., Long K., Agnihothram S., Whitmore A., Ferris M., Funkhouser W., Gralinski L., Totura A., Heise M., et al. A Double-Inactivated Severe Acute Respiratory Syndrome Coronavirus Vaccine Provides Incomplete Protection in Mice and Induces Increased Eosinophilic Proinflammatory Pulmonary Response upon Challenge//J. Virol. - 2011. - V. 85-№23. -P. 12201-15.
3. Jaume M., Yip M.S., Kam Y.W., Cheung C.Y., Kien F., Roberts A., Li P.H., Dutry I., Escriou N., Daeron M., et al. SARS CoV subunit vaccine: antibody-mediated neutralisation and enhancement.//Hong Kong Med. J. = Xianggang yi xue zazhi. - 2012. - V. 18Suppl2 - №1. - P. 31-6.
4. Xia S., Yan L., Xu W., Agrawal A.S., Algaissi A., Tseng C.T.K., Wang Q., Du L., Tan W., Wilson I.A., et al. A pan-coronavirus fusion inhibitor targeting the HR1 domain of human coronavirus spike//Sci. Adv. - 2019. - V. 5 - №4. - P 45-34.
5. Gui M., Song W., Zhou H., Xu J., Chen S., Xiang Y., Wang X. Cryo-electron microscopy structures of the SARS-CoV spike glycoprotein reveal a prerequisite conformational state for receptor binding//Cell Res. - 2017. - V. 27 - №1. - P. 119-29.
6. He Y., Zhou Y., Liu S., Kou Z., Li W., Farzan M., Jiang S. Receptor-binding domain of SARS-CoV spike protein induces highly potent neutralizing antibodies: Implication for developing subunit vaccine//Biochem. Biophys. Res. Commun. - 2004. - V. 324 - №2. - P. 773-81.
7. Du L., Zhao G., Yang Y., Qiu H., Wang L., Kou Z., Tao X., Yu H., Sun S., Tseng C.T.K., et al. A Conformation-Dependent Neutralizing Monoclonal Antibody Specifically Targeting Receptor-Binding Domain in Middle East Respiratory Syndrome Coronavirus Spike Protein//J. Virol. - 2014. - V. 88 - №12. - P. 7045-53.
8. Walls A.C., Xiong X., Park Y.J., Tortorici M.A., Snijder J., Quispe J., Cameroni E., Gopal R., Dai M., Lanzavecchia A., et al. Unexpected Receptor Functional Mimicry Elucidates Activation of Coronavirus Fusion//Cell. - 2019. - V. 176 - №5. - P. 1026-1039.el5.
9. Chen W.H., Chag S.M., Poongavanam M.V., Biter A.B., Ewere E.A., Rezende W., Seid C.A., Hudspeth E.M., Pollet J., McAtee C.P., et al. Optimization of the Production Process and Characterization of the Yeast-Expressed SARS-CoV Recombinant Receptor-Binding Domain (RBD219-N1), a SARS Vaccine Candidate//J. Pharm. Sci. - 2017. - V. 106 - №8. - P. 1961-70.
10. Zang J., Gu C, Zhou В., Zhang C, Yang Y., Xu S., Bai L., Zhang R., Deng Q., Yuan Z., et al. Immunization with the receptor-binding domain of SARS-CoV-2 elicits antibodies cross-neutralizing SARS-CoV-2 and SARS-CoV without antibody-dependent enhancement//Cell Discov. - 2020. - V. 6 - №1. - P. 4-7.
11. Yang J., Wang W., Chen Z., Lu S., Yang F., Bi Z., Bao L., Mo F., Li X., Huang Y., et al. A vaccine targeting the RBD of the S protein of SARS-CoV-2 induces protective immunity//Nature. - 2020. - V. 586 - №7830. - P. 572-7.
12. Malladi S.K., Singh R., Pandey S., Gayathri S., Kanjo K., Ahmed S., Khan M.S., Kalita P., Girish N., Upadhyaya A., et al. Design of a highly thermotolerant, immunogenic SARS-CoV-2 spike fragment//J. Biol. Chem. - 2021. -V. 296100025.
13. Маниатис Т., Фрич Э, Сэмбрук Дж. Молекулярное клонирование, М.: Мир, 1984; Клонирование ДНК. Методы. Под ред. Д.Гловера, Пер. с англ., Москва, Мир, 1988; Saiki R.K. et al. Science. 1988, 239(4839):487-491; Sanger F. et al. Proc. Nat. Acad. Sci. 1977, 74:5463-5467.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3"
fileName="RBDdelta_05.07.2023.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-07-05">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1234567</ApplicationNumberText>
<FilingDate>2023-07-05</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1234567</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1234567</ApplicationNumberText>
<FilingDate>2023-07-05</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Государственный научный центр вирусологии и биотехнологии
«Вектор» Федеральной службы по надзору в сфере защиты прав
потребителей и благополучия человека (ФБУН ГНЦ ВБ «Вектор»
Роспотребнадзора)</ApplicantName>
<ApplicantNameLatin>Federalnoe byudzhetnoe uchrezhdenie nauki
"Gosudarstvennyj nauchnyj tsentr virusologii i biotekhnologii
"Vektor" Federalnoj sluzhby po nadzoru v sfere zashchity
prav potrebitelej i blagopoluchiya cheloveka (FBUN GNTS VB
"Vektor" Rospotrebnadzora) (RU)</ApplicantNameLatin>
<InventionTitle languageCode="ru">Интегративный плазмидный вектор
pVEAL3- RBDdel, обеспечивающий синтез и секрецию рекомбинантного
рецепторсвязывающего домена RBDdelta коронавируса SARS-CoV-2 в
клетках млекопитающих, рекомбинантный штамм клеточной линии CHO-K1-
RBDdelta и рекомбинантный белок RBDdelta SARS-CoV-2, продуцируемый
указанным штаммом клеточной линии </InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>5160</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5160</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgaagaaaggcccacccgtgaaggtgagccagtgagttgattgcagtcc
agttacgctggagtctgaggctcgtcctgaatggatccctatacagttgaagtcggaagtttacatacac
ttaagttggagtcattaaaactcgtttttcaactactccacaaatttcttgttaacaaacaatagttttg
gcaagtcagttaggacatctactttgtgcatgacacaagtcatttttccaacaattgtttacagacagat
tatttcacttataattcactgtatcacaattccagtgggtcagaagtttacatacactaagttcgactcc
tctgcagaatgcggcgatgtttcggtaaggggtccgctactagttattaatagtaatcaattacggggtc
attagttcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctgaccg
cccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttcc
attgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgacctta
tgggactttcctacttggcagtacatctacgtattagtcatcgctattaccatggtgatgcggttttggc
agtacatcaatgggcgtggatagcggtttgactcacggggatttccaagtctccaccccattgacgtcaa
tgggagtttgttttggcaccaaaatcaacgggactttccaaaatgtcgtaacaactccgccccattgacg
caaatgggcggtaggcgtgtacggtgggaggtctatataagcagagctctctggctaactagagaaccca
ctgcttactggcttatcgaaattaatacgactcactatagggagacccaagctggctagccatgtttgtt
tttcttgttttattgccactagtctctagtcagtgtgtggaaaagggcatctaccagaccagcaacttcc
gggtgcagcccaccgaatccatcgtgcggttccccaatatcaccaatctgtgccccttcggcgaggtgtt
caatgccaccagattcgcctctgtgtacgcctggaaccggaagcggatcagcaattgcgtggccgactac
tccgtgctgtacaactccgccagcttcagcaccttcaagtgctacggcgtgtcccctaccaagctgaacg
acctgtgcttcacaaacgtgtacgccgacagcttcgtgatccggggagatgaagtgcggcagattgcccc
tggacagacaggcaagatcgccgactacaactacaagctgcccgacgacttcaccggctgtgtgattgcc
tggaacagcaacaacctggactccaaagtcggcggcaactacaattaccggtaccggctgttccggaagt
ccaatctgaagcccttcgagcgggacatctccaccgagatctatcaggccggcagcaagccttgtaacgg
cgtggaaggcttcaactgctacttcccactgcagtcctacggctttcagcccacaaatggcgtgggctat
cagccctacagagtggtggtgctgagcttcgaactgctgcatgcccctgccacagtgtgcggccctaaga
aaagcaccaatctcgtgaagaacaaatgcgtgaacttccatcaccaccatcatcaccatcaccaccacta
agcttaagtttaaaccgctgatcagcctcgtcgaccgagcggttcccgcccctctccctcccccccccct
aacgttactggccgaagccgcttggaataaggccggtgtgcgtttgtctatatgttattttccaccatat
tgccgtcttttggcaatgtgagggcccggaaacctggccctgtcttcttgacgagcattcctaggggtct
ttcccctctcgccaaaggaatgcaaggtctgttgaatgtcgtgaaggaagcagttcctctggaagcttct
tgaagacaaacaacgtctgtagcgaccctttgcaggcagcggaaccccccacctggcgacaggtgcctct
gcggccaaaagccacgtgtataagatacacctgcaaaggcggcacaaccccagtgccacgttgtgagttg
gatagttgtggaaagagtcaaatggctcacctcaagcgtattcaacaaggggctgaaggatgcccagaag
gtaccccattgtatgggatctgatctggggcctcggtgcacatgctttacatgtgtttagtcgaggttaa
aaaacgtctaggccccccgaaccacggggacgtggttttcctttgaaaaacacgatgataatatggccac
aaccatgaccgagtacaagcccacggtgcgcctcgccacccgcgacgacgtccccagggccgtacgcacc
ctcgccgccgcgttcgccgactaccccgccacgcgccacaccgtcgatccggaccgccacatcgagcggg
tcaccgagctgcaagaactcttcctcacgcgcgtcgggctcgacatcggcaaggtgtgggtcgcggacga
cggcgccgcggtggcggtctggaccacgccggagagcgtcgaagcgggggcggtgttcgccgagatcggc
ccgcgcatggccgagttgagcggttcccggctggccgcgcagcaacagatggaaggcctcctggcgccgc
accggcccaaggagcccgcgtggttcctggccaccgtcggcgtctcgcccgaccaccagggcaagggtct
gggcagcgccgtcgtgctccccggagtggaggcggccgagcgcgccggggtgcccgccttcctggagacc
tccgcgccccgcaacctccccttctacgagcggctcggcttcaccgtcaccgccgacgtcgaggtgcccg
aaggaccgcgcacctggtgcatgacccgcaagcccggtgcctgattcgcatatgggttaatgcttcgagc
agacatgataagatacattgatgagtttggacaaaccacaactagaatgcagtgaaaaaaatgctttatt
tgtgaaatttgtgatgctattgctttatttgtaaccattataagctgcaataaacaagttcctcgacctc
tagctagagctactcgggaccccttaccgaaacatcgccgcattctgcagaggagtcgagtgtatgtaaa
cttctgacccactgggaatgtgatgaaagaaataaaagctgaaatgaatcattctctctactattattct
gatatttcacattcttaaaataaagtggtgatcctaactgacctaagacagggaatttttactaggatta
aatgtcaggaattgtgaaaaagtgagtttaaatgtatttggctaaggtgtatgtaaacttccgacttcaa
ctgtatagggatccgctcaatactgaccatttaaatcatacctgacctccatagcagaaagtcaaaagcc
tccgaccggaggcttttgacttgatcggcacgtaagaggttccaactttcaccataatgaaataagatca
ctaccgggcgtattttttgagttatcgagattttcaggagctaaggaagctaaaatgagccatattcaac
gggaaacgtcttgctcgaggccgcgattaaattccaacatggatgctgatttatatgggtataaatgggc
tcgcgataatgtcgggcaatcaggtgcgacaatctatcgattgtatgggaagcccgatgcgccagagttg
tttctgaaacatggcaaaggtagcgttgccaatgatgttacagatgagatggtcaggctaaactggctga
cggaatttatgcctcttccgaccatcaagcattttatccgtactcctgatgatgcatggttactcaccac
tgcgatcccagggaaaacagcattccaggtattagaagaatatcctgattcaggtgaaaatattgttgat
gcgctggcagtgttcctgcgccggttgcattcgattcctgtttgtaattgtccttttaacggcgatcgcg
tatttcgtctggctcaggcgcaatcacgaatgaataacggtttggttggtgcgagtgattttgatgacga
gcgtaatggctggcctgttgaacaagtctggaaagaaatgcataagcttttgccattctcaccggattca
gtcgtcactcatggtgatttctcacttgataaccttatttttgacgaggggaaattaataggttgtattg
atgttggacgagtcggaatcgcagaccgataccaggatcttgccatcctatggaactgcctcggtgagtt
ttctccttcattacagaaacggctttttcaaaaatatggtattgataatcctgatatgaataaattgcag
tttcacttgatgctcgatgagtttttctaatgagggcccaaatgtaatcacctggctcaccttcgggtgg
gcctttctgcgttgctggcgtttttccataggctccgcccccctgacgagcatcacaaaaatcgatgctc
aagtcagaggtggcgaaacccgacaggactataaagataccaggcgtttccccctggaagctccctcgtg
cgctctcctgttccgaccctgccgcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgc
tttctcatagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgca
cgaaccccccgttcagcccgaccgctgcgccttatccggtaactatcgtcttgagtccaacccggtaaga
cacgacttatcgccactggcagcagccactggtaacaggattagcagagcgaggtatgtaggcggtgcta
cagagttcttgaagtggtggcctaactacggctacactagaagaacagtatttggtatctgcgctctgct
gaagccagttacctcggaaaaagagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtg
gtttttttgtttgcaagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgattttcta
c</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>732</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..732</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtggaaaagggcatctaccagaccagcaacttccgggtgcagcccaccg
aatccatcgtgcggttccccaatatcaccaatctgtgccccttcggcgaggtgttcaatgccaccagatt
cgcctctgtgtacgcctggaaccggaagcggatcagcaattgcgtggccgactactccgtgctgtacaac
tccgccagcttcagcaccttcaagtgctacggcgtgtcccctaccaagctgaacgacctgtgcttcacaa
acgtgtacgccgacagcttcgtgatccggggagatgaagtgcggcagattgcccctggacagacaggcaa
gatcgccgactacaactacaagctgcccgacgacttcaccggctgtgtgattgcctggaacagcaacaac
ctggactccaaagtcggcggcaactacaattaccggtaccggctgttccggaagtccaatctgaagccct
tcgagcgggacatctccaccgagatctatcaggccggcagcaagccttgtaacggcgtggaaggcttcaa
ctgctacttcccactgcagtcctacggctttcagcccacaaatggcgtgggctatcagccctacagagtg
gtggtgctgagcttcgaactgctgcatgcccctgccacagtgtgcggccctaagaaaagcaccaatctcg
tgaagaacaaatgcgtgaacttccatcaccaccatcatcaccatcaccaccac</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>215</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..215</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRK
RISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLP
DDFTGCVIAWNSNNLDSKVGGNYNYRYRLFRKSNLKPFERDISTEIYQAGSKPCNGVEGFNCYFPLQSYG
FQPTNGVGYQPYRVVVHHHHHHHHHH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---




Изобретение относится к биотехнологии. Описан интегративный плазмидный вектор pVEAL3-RBDdelta, обеспечивающий синтез и секрецию белка RBDdelta коронавируса SARS-CoV-2 в клетках млекопитающих. Вектор имеет размер 5160 п.н., нуклеотидную последовательность SEQ ID NO: 1 и включает кодон-оптимизированную нуклеотидную последовательность SEQ ID NO: 2 гена, кодирующего RBDdelta коронавируса SARS-CoV-2, в которую внесены 2 мутации для получения белка RBD варианта В. 1.617.2. Раскрыт рекомбинантный штамм клеточной линии яичника китайского хомячка СНО-K1-RBDdelta продуцент рекомбинантного белка рецепторсвязывающего домена RBDdelta коронавируса SARS-CoV-2. Указанный штамм содержит интегративный плазмидный вектор pVEAL3 - RBDdelta и депонирован под номером 327 в коллекции культур клеток ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора. Также описан рекомбинантный белок RBDdelta коронавируса SARS-CoV-2, который продуцируется клеточной линией яичника китайского хомячка СНО-K1-RBDdelta, имеет аминокислотную последовательность SEQ ID NO: 3 и предназначен для создания иммунобиологических препаратов. Изобретение направлено на повышение выхода целевого рекомбинантного белка RBDdelta SARS-CoV-2. 3 н.п. ф-лы, 7 ил., 1 табл., 4 пр.
1. Интегративный плазмидный вектор pVEAL3-RBDdelta, обеспечивающий синтез и секрецию белка RBDdelta коронавируса SARS-CoV-2 в клетках млекопитающих, имеющий размер 5160 п.н., нуклеотидную последовательность SEQ ID NO: 1 и содержащий в соответствии с физической и генетической картой, представленной на фиг. 1, следующие конструктивные элементы:
- участок начала репликации ori, имеющий координаты с 4552 по 5139;
- последовательности прямых и инвертированных повторов IR/DR, содержащие сайты связывания с транспозазой SB 100, имеющие координаты с 88 по 368 и с 3210 по 3487;
- CMV энхансер, имеющий координаты с 369 по 733, и CMV промотор, имеющий координаты с 734 по 937 и являющийся наиболее часто используемым промотором в генно-терапевтических конструкциях;
- лидерный пептид S-SP, обеспечивающий экспорт белка из клеток СНО-K1-RBDdelta и имеющий координаты с 3090 по 3134;
- кодон-оптимизированная нуклеотидная последовательность SEQ ID NO: 2 гена, кодирующего рецепторсвязывающий домен RBDdelta коронавируса SARS-CoV-2, в которую внесены 2 мутации для получения белка RBD варианта В. 1.617.2 и которая имеет координаты с 1066 по 1767;
- последовательность 10xHis, имеющая координаты с 1768 по 1797 и являющаяся полигистидиновым тэгом для очистки рекомбинантного белка с помощью металл-хеллатной хроматографии;
- участок внутренней посадки рибосомы EMCV IRES, имеющий координаты с 1847 по 2421;
- последовательность PuroR, кодирующая фактор устойчивости к антибиотику пуромицину, имеющая координаты с 2434 по 3033;
- последовательность SV40 poly(A) signal для стабилизации мРНК-транскриптов за счет полиаденилирования, имеющая координаты с 3068 по 3189;
- промотор cat синтетический бактериальный промотор, имеющий координаты с 3571 по 3673;
- нуклеотидная последовательность KanR, кодирующая фактор устойчивости к антибиотику канамицину, позволяющая проводить амплификацию плазмиды в E.coli и имеющая координаты с 3674 по 4489.
2. Рекомбинантный штамм клеточной линии яичника китайского хомячка СНО-K1-RBDdelta - продуцент рекомбинантного белка рецепторсвязывающего домена RBDdelta коронавируса SARS-CoV-2, содержащий интегративный плазмидный вектор pVEAL3-RBDdelta по п. 1, и депонированный под номером 327 в коллекции культур клеток ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора.
3. Рекомбинантный белок RBDdelta коронавируса SARS-CoV-2, продуцируемый рекомбинантным штаммом клеточной линии яичника китайского хомячка СНО-K1-RBDdelta по п. 2, имеющий аминокислотную последовательность SEQ ID NO: 3 и предназначенный для создания иммунобиологических препаратов.
| Интегративный плазмидный вектор pVEAL2-S-RBD, обеспечивающий экспрессию и секрецию рекомбинантного рецепторсвязывающего домена (RBD) коронавируса SARS-CoV-2 в клетках млекопитающих, рекомбинантный штамм клеточной линии CHO-K1-RBD и рекомбинантный белок RBD SARS-CoV-2, продуцируемый указанным штаммом клеточной линии CHO-K1-RBD | 2021 |
|
RU2752858C1 |
| Тележка для перевозки безгребневых колесных пар | 1933 |
|
SU35233A1 |
| WO 2022045935 A1, 03.03.2022. | |||
Авторы
Даты
2024-03-26—Публикация
2023-07-28—Подача