Перекрестная ссылка на родственные заявки
[01] Настоящая заявка испрашивает приоритет на основании предварительной заявки на патент США №62/753558, поданной 31 октября 2018 г., содержание которой во всей полноте включено в настоящую заявку посредством ссылки.
Перечень последовательностей
[02] Настоящая заявка содержит Перечень последовательностей, представленный в электронном виде посредством EFS-Web в Ведомство по патентам и товарным знакам США в виде текстового файла ASCII под названием «IP-1546-PCT_ST25.txt» с размером 224 килобайта и созданного 31 октября 2019 г. Информация, содержащаяся в Перечне последовательностей, включена в настоящую заявку посредством ссылки.
Область изобретения
[03] Настоящее раскрытие относится, среди прочего, к измененным полимеразам для применения при проведении реакции включения нуклеотидов, особенно в контексте секвенирования нуклеиновых кислот путем синтеза.
Уровень техники изобретения
[04] Технология секвенирования следующего поколения (NGS, от англ. next-generation sequencing) основана на ДНК-полимеразах в качестве важнейшего компонента процесса секвенирования. Сокращение времени на секвенирование матрицы при сохранении высокой достоверности является желательным. Сокращение каждого цикла процесса секвенирования путем синтеза (SBS, от англ. sequencing by synthesis) является эффективным шагом для достижения более короткого времени прогона секвенирования. Один из подходов к сокращению времени цикла состоит в сокращении времени этапа включения. Тем не менее, хотя сокращение времени включения может значительно улучшить общее время прогона, обычно это происходит в ущерб достоверности. Например, частота фазирования, частота предварительного фазирования и/или частота разветвления (bypass) увеличиваются, и, как следствие, увеличивается частота ошибок. При низкой частоте ошибок во время прогона секвенирования большинство молекул матрицы в кластере оканчиваются одним и тем же меченым нуклеотидом, и сигнал является четким. Напротив, при пониженной достоверности во время прогона секвенирования все большее количество молекул матрицы в кластере оканчиваются неверно меченым нуклеотидом, и сигнал может иметь повышенный уровень шума для точного определения, какой нуклеотид был включен.
Описание изобретения
[05] В настоящей заявке предложены рекомбинантные ДНК-полимеразы. Один пример полимеразы согласно настоящему раскрытию включает аминокислотную последовательность, которая по меньшей мере на 80% идентична аминокислотной последовательности SEQ ID NO: 1 ДНК-полимеразы 9°N.
[06] В одном варианте реализации полимераза также включает мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, и по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Phe152, Val278, Met329, Val471, Thr514, Leu631 или Glu734 в аминокислотной последовательности ДНК-полимеразы 9°N, и необязательно дополнительно включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[07] В одном варианте реализации полимераза также включает мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, и по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Lys476, Lys477, Thr514, IIe521 или Thr590 в аминокислотной последовательности ДНК-полимеразы 9°N, и необязательно дополнительно включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[08] В одном варианте реализации полимераза также включает мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, и по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Arg247, Glu599, Lys620, His633 или Val661 в аминокислотной последовательности ДНК-полимеразы 9°N, и необязательно дополнительно включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[09] В одном варианте реализации полимераза также включает (i) мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, (ii) по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Phe152, Val278, Met329, Val471, Leu631 или Glu734 в аминокислотной последовательности ДНК-полимеразы 9°N, и (Hi) по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Lys476, Lys477, Thr514, IIe521 или Thr590 в аминокислотной последовательности ДНК-полимеразы 9°N, и необязательно дополнительно включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[010] В одном варианте реализации полимераза также включает (i) мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, (ii) по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Lys476, Lys477, Thr514, IIe521 или Thr590 в аминокислотной последовательности ДНК-полимеразы 9°N, и (iii) по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Arg247, Glu599, Lys620, His633 или Val661 в аминокислотной последовательности ДНК-полимеразы 9°N, и необязательно дополнительно включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[011] В одном варианте реализации полимераза также включает (i) мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, (ii) по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Phe152, Val278, Met329, Val471, Leu631 или Glu734 в аминокислотной последовательности ДНК-полимеразы 9°N, (iii) по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Lys476, Lys477, Thr514, Ile521 или Thr590 в аминокислотной последовательности ДНК-полимеразы 9°N, и (iv) по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Arg247, Glu599, Lys620, His633 или Val661 в аминокислотной последовательности ДНК-полимеразы 9°N, и необязательно дополнительно включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[012] Другой пример полимеразы согласно настоящему раскрытию включает аминокислотную последовательность, которая по меньшей мере на 80% идентична аминокислотной последовательности SEQ ID NO: 8 ДНК-полимеразы 9°N, и также включает (i) мутации с заменой аминокислоты в положениях, функционально эквивалентных Tyr497, Phe152, Val278, Met329, Val471 и Thr514 в аминокислотной последовательности ДНК-полимеразы 9°N; (ii) мутации с заменой аминокислоты в положениях, функционально эквивалентных Tyr497, Met329, Val471 и Glu734 в аминокислотной последовательности ДНК-полимеразы 9°; (iii) мутации с заменой аминокислоты в положениях, функционально эквивалентных Tyr497, Arg247, Glu599 и His633 в аминокислотной последовательности ДНК-полимеразы 9°N; (iv) мутации с заменой аминокислоты в положениях, функционально эквивалентных Tyr497, Arg247, Glu599, Lys620 и His633 в аминокислотной последовательности ДНК-полимеразы 9°N; (v) мутации с заменой аминокислоты в положениях, функционально эквивалентных Tyr497, Met 329, Thr514, Lys620 и Val661 в аминокислотной последовательности ДНК-полимеразы 9°N; или (vi) мутации с заменой аминокислоты в положениях, функционально эквивалентных Tyr497, Val278, Val471, Arg247, Glu599 и His633 в аминокислотной последовательности ДНК-полимеразы 9°N.
[013] В настоящей заявке также предложена рекомбинантная ДНК-полимераза, которая включает аминокислотную последовательность согласно любой из SEQ ID NO: 10-34, молекула нуклеиновой кислоты, которая кодирует раскрытую в настоящей заявке полимеразу, вектор экспрессии, который включает молекулу нуклеиновой кислоты, и клетка-хозяин, которая включает указанный вектор.
[014] Настоящее раскрытие также включает способы. В одном варианте реализации способ предназначен для включения модифицированных нуклеотидов в растущую цепь ДНК. Способ включает обеспечение взаимодействия следующих компонентов: (i) полимеразы, описанной в настоящей заявке; (ii) матрицы ДНК; и (iii) раствора нуклеотидов.
[015] В настоящей заявке также предложен набор. В одном варианте реализации набор предназначен для проведения реакции включения нуклеотидов. Набор может включать, например, описанную в настоящей заявке полимеразу и раствор нуклеотидов.
Краткое описание графических материалов
[016] На ФИГ. 1 схематически показано выравнивание аминокислотных последовательностей полимеразы из Thermococcus sp.9°N-7 (9°N, SEQ ID NO: 1), Thermococcus litoralis (Vent, SEQ ID NO: 2 и Deep Vent, SEQ ID NO: 3), Thermococcus waiotapuensis (Twa, SEQ ID NO: 7), Thermococcus kodakaraenis (KOD, SEQ ID NO: 5), Pyrococcus furiosus (Pfu, SEQ ID NO: 4), Pyrococcus abyssi (Pab, SEQ ID NO: 6). «*» (Звездочка) указывает положения, которые имеют один полностью консервативный остаток между всеми полимеразами. «:» (Двоеточие) указывает на сохранение сильно схожих свойств между группами, как показано ниже, что примерно эквивалентно оценке больше 0,5 в матрице Gonnet РАМ 250. "." (Точка) указывает на сохранение слабо схожих свойств между группами, как показано ниже, что примерно эквивалентно оценке меньше или равно 0,5 и больше 0 в матрице Gonnet РАМ 250.
[017] На ФИГ. 2 показано снижение частоты ошибок фазирования и накопленных ошибок при коротком времени включения, продемонстрированное одной из измененных полимераз согласно настоящему раскрытию, Pol 1558 (SEQ ID NO: 11), по сравнению с контролем Pol 812 (SEQ ID NO: 8) (панели слева). Два фермента показывают сравнимую частоту фазирования и ошибок при стандартном времени включения (панели справа).
[018] На ФИГ. 3А показано снижение частоты ошибок фазирования R1 и накопленных ошибок Е. coli при коротком времени включения, продемонстрированное выбранными измененными полимеразами согласно настоящему раскрытию, Pol 1558 (SEQ ID NO: 11), Pol 1671 (SEQ ID NO: 23), Pol 1682 (SEQ ID NO: 25) и Pol 1745 (SEQ ID NO: 28), no сравнению с контролями Pol 812 (SEQ ID NO: 8) и Pol 963 (SEQ ID NO: 9). Пунктирные линии на верхней и нижней панелях показывают накопленную ошибку Е. coli и частоту фазирования R1, продемонстрированные Pol 812 при стандартном времени включения. На ФИГ. 3В представлено сравнение частоты фазирования и предварительного фазирования тех же измененных полимераз относительно контролей Pol 812 и Pol 963.
[019] На ФИГ. 4 представлено сравнение частоты накопленных ошибок PhiX для Pol 1550 (SEQ ID NO: 10) и Pol 1558 (SEQ ID NO: 11) с таковой для контроля Pol 812 (SEQ ID NO: 8) при стандартном и коротком временах включения во время длинных прочтений секвенирования (2×250 циклов). Оба мутанта демонстрируют заметное снижение частоты ошибок после секвенирования спаренных концов.
[020] На ФИГ. 5А показано сравнение показателей секвенирования NovaSeq™ для одной из измененных полимераз согласно настоящему изобретению, Pol 1671 (SEQ ID NO: 23), продемонстрированных при коротких временах включения, с таковыми для контроля Pol 812 (SEQ ID NO: 8), продемонстрированных при стандартном и коротком времени включения. Верхние панели демонстрируют процент кластеров, прошедших фильтр («Clusters PF»); нижние панели демонстрируют частоту накопленных ошибок PhiX. Светлые незаштрихованные кружки обозначают показатели Pol 812 при стандартном времени включения, тогда как темные незаштрихованные кружки обозначают показатели Pol 812 при коротком времени включения. Все показатели Pol 1671, обозначенные заштрихованными кружками, имеют короткое время включения. На ФИГ. 5 В обобщены частоты накопленных ошибок PhiX, значения Q30 и частоты фазирования, которые демонстрирует Pol 1671 по сравнению с контролем Pol 812 для прочтений 1 и 2 NovaSeq™ при стандартном и коротком времени включения. Значительное улучшение качества для обоих прочтений наблюдалось при применении Pol 1671.
Подробное описание изобретения
[021] Термин «и/или» обозначает один или все перечисленные элементы или комбинацию любых двух или более из перечисленных элементов.
[022] Слова «предпочтительный» и «предпочтительно» относятся к вариантам реализации изобретения, которые могут обеспечить определенные преимущества при определенных обстоятельствах. Однако другие варианты реализации также могут быть предпочтительными при тех же или других обстоятельствах. Кроме того, перечисление одного или более предпочтительных вариантов реализации не подразумевает, что другие варианты реализации не являются подходящими, и не предназначено для исключения других вариантов реализации из объема настоящего изобретения.
[023] Термины «содержит» и их варианты не имеют ограничивающего значения там, где эти термины появляются в описании и формуле изобретения.
[024] Следует понимать, что везде, где варианты реализации описаны в настоящей заявке с применением формулировки «включать», «включает» или «включая» и тому подобных, в остальном аналогичные варианты реализации также описаны терминами «состоящий из» и/или «состоящий по существу из».
[025] Если не указано иное, определенные и неопределенные формы единственного числа и «по меньшей мере один» используются взаимозаменяемо и обозначают один или более.
[026] Условия, которые «подходят» для возникновения события, или «подходящие» условия представляют собой условия, которые не препятствуют возникновению таких событий. Таким образом, данные условия позволяют, усиливают, облегчают и/или способствуют осуществлению события.
[027] В настоящей заявке «обеспечение» в контексте композиции, изделия, нуклеиновой кислоты или ядра обозначает получение композиции, изделия, нуклеиновой кислоты или ядра, приобретение композиции, изделия, нуклеиновой кислоты или ядра или получение другим способом соединения, композиции, изделия или ядра.
[028] Также в настоящей заявке перечисление числовых диапазонов по конечным точкам включает все числа, входящие в этот диапазон (например, от 1 до 5 включает 1, 1,5, 2, 2,75, 3, 3,80, 4, 5 и т.д.).
[029] В настоящей заявке ссылка на «один вариант реализации», «вариант реализации», «определенные варианты реализации» или «некоторые варианты реализации» и т.д. обозначает, что конкретный признак, конфигурация, композиция или характеристика, описанные в связи с указанным вариантом реализации, включены по меньшей мере в один вариант реализации согласно настоящему раскрытию. Таким образом, появление таких фраз в различных местах в настоящей заявке не обязательно относится к одному и тому же варианту реализации согласно настоящему раскрытию. Кроме того, конкретные признаки, конфигурации, композиции или характеристики могут быть объединены любым подходящим образом в одном или более вариантах реализации.
[030] Поддержанию или превышению текущих уровней эффективности при более быстром времени включения может способствовать новое поколение полимераз. В настоящей заявке представлены полимеразные ферменты, обладающие в значительной степени улучшенной эффективностью в условиях быстрого цикла секвенирования путем синтеза (SBS). Авторы настоящего изобретения неожиданно идентифицировали некоторые измененные полимеразы, которые демонстрируют улучшенные характеристики, включая повышенную точность в течение короткого времени включения. Повышенная точность включает снижение частоты ошибок и снижение фазирования. Измененные полимеразы имеют ряд других связанных преимуществ, в том числе более низкое предварительное фазирование, более низкую частоту разветвления и улучшенные показатели качества в реакциях SBS. Это улучшение сохраняется даже при применении полимеразы в более низких концентрациях. Соответственно, в одном варианте реализации концентрация ДНК-полимеразы в реакции SBS может составлять от 120 нг/мкл до 80 нг/мкл. В одном варианте реализации концентрация ДНК-полимеразы в реакции SBS может составлять не более 120 нг/мкл, не более 110 нг/мкл, не более 100 нг/мкл или не более 90 нг/мкл. В одном варианте реализации концентрация ДНК-полимеразы в реакции SBS может составлять по меньшей мере 80 нг/мкл, по меньшей мере 90 нг/мкл, по меньшей мере 100 нг/мкл или по меньшей мере 110 нг/мкл.
[031] Частота ошибок относится к измерению частоты встречаемости ошибок при идентификации правильного основания, то есть комплементарной цепи к последовательности матрицы в конкретном положении, во время реакции секвенирования. Достоверность, с которой секвенированная библиотека соответствует исходной последовательности генома, может варьироваться в зависимости от частоты встречаемости мутаций оснований, возникающих на любом этапе от экстракции нуклеиновой кислоты до ее секвенирования на платформе для секвенирования. Эта частота встречаемости устанавливает верхний предел вероятности правильности секвенированного основания. В некоторых вариантах реализации показатель качества представлен в виде числового значения. Например, показатель качества может быть указан как QXX, где XX представляет собой показатель, и он означает, что данный конкретный запрос имеет вероятность ошибки 10-XX/10. Таким образом, например, Q30 соответствует частоте ошибок 1 из 1000 или 0,1%, а Q40 соответствует частоте ошибок 1 из 10000 или 0,01%.
[032] Фазирование и предварительное фазирование представляют собой термины, известные специалистам в данной области техники, и они используются для описания утраты синхронности при прочтении копий последовательности кластера. Фазирование и предварительное фазирование обусловливают включение сигнала текущего цикла и шума из предыдущего и последующего циклов в выбранные интенсивности определенного цикла. Таким образом, в настоящей заявке термин «фазирование» относится к явлению в SBS, которое вызвано неполным включением нуклеотида в некоторую часть цепей ДНК внутри кластеров полимеразами в данном цикле секвенирования, и, таким образом, является мерой частоты, с которой отдельные молекулы в кластере утрачивают синхронность друг с другом. Фазирование может быть измерено во время детектирования сигнала кластера в каждом цикле и может быть представлено как процент детектируемого сигнала от кластера, который не является синхронным сигналу в кластере. Например, кластер детектируется по «зеленому» сигналу флуорофора во время цикла N. В последующем цикле (цикл N плюс 1) 99,9% сигнала кластера детектируется в «красном» канале, и 0,1% сигнала остается от предыдущего цикла и детектируется в «зеленом» канале. Данный результат будет указывать на возникновение фазирования, и может быть представлен в виде числового значения, такого как значение фазирования 0,1, указывающее, что 0,1% молекул в кластере смещены назад в каждом цикле.
[033] В настоящей заявке термин «предварительное фазирование» относится к явлению в SBS, которое вызвано включением нуклеотидов без эффективных 3'-терминаторов, вследствие чего событие включения происходит на один цикл вперед. По мере увеличения числа циклов доля последовательностей в кластере, на которые влияет фазирование, увеличивается, что затрудняет идентификацию правильного основания. Предварительное фазирование можно детектировать прибором для секвенирования и представить в виде числового значения, такого как значение предварительного фазирования 0,1, что указывает на то, что 0,1% молекул в кластере продвинулись вперед в каждом цикле.
[034] Детектирование фазирования и предварительного фазирования может быть осуществлено и зарегистрировано в соответствии с любой подходящей методологией, известной в данной области техники, например, как описано в патенте США №8965076. Например, как описано в приведенных ниже примерах, фазирование детектируют и регулярно регистрируют во время прогонов секвенирования SBS на таких приборах для секвенирования, как платформы для секвенирования HiSeq™, Genome Analyzer™, NextSeq™, NovaSeq™, iSeq™, MiniSeq™ или MiSeq™ от IIIumina, Inc. (Сан-Диего, Калифорния) или на любом другом подходящем приборе, известном в данной области техники.
[035] Уменьшение времени цикла может увеличивать частоту возникновения фазирования, предварительного фазирования и/или разветвления, каждая из которых вносит свой вклад в частоту ошибок. Обнаружение измененных полимераз, которые уменьшают частоту фазирования, предварительного фазирования и/или разветвления, даже при применении в условиях быстрого цикла, является неожиданным и обеспечивает большое преимущество при применении в SBS. Например, измененные полимеразы могут обеспечить более быстрое время цикла SBS, более низкие значения фазирования и предварительного фазирования и/или большую длину прочтения секвенирования. Характеристики частоты ошибок и фазирования для измененных полимераз, как предложено в настоящей заявке, изложены в разделе «Примеры» ниже.
Полимеразы
[036] В настоящей заявке предложены полимеразы, композиции, включающие полимеразу, и способы применения полимеразы. Раскрытая в настоящей заявке полимераза представляет собой ДНК-полимеразу. В одном варианте реализации полимераза согласно настоящему раскрытию, также называемая в настоящей заявке «измененной полимеразой», основана на аминокислотной последовательности эталонной полимеразы. Измененная полимераза включает мутации по типу замены в одном или более остатках по сравнению с эталонной полимеразой. Мутация по типу замены может находиться в том же положении или в функционально эквивалентном положении по сравнению с эталонной полимеразой. Эталонные полимеразы и функционально эквивалентные положения подробно описаны в настоящей заявке. Специалисту в данной области техники будет легко понятно, что описанная в настоящей заявке измененная полимераза не встречается в природе.
[037] Раскрытая в настоящей заявке эталонная полимераза имеет частоту ошибок, подходящую для SBS реакций; однако применение эталонной полимеразы в SBS реакциях с более коротким временем включения увеличивает частоту ошибок. Измененная полимераза, описанная в настоящей заявке, поддерживает частоты ошибок, превосходящие наблюдаемые частоты для эталонных полимераз, даже когда измененную полимеразу применяют в SBS реакциях с более коротким временем включения. В одном варианте реализации уменьшенные частоты ошибок возникают, когда измененную полимеразу тестируют с применением быстрого времени включения. Включение относится к количеству времени, в течение которого ДНК-полимераза находится в контакте с матрицей. В настоящей заявке медленное время включения представляет собой время включения, применяемое в стандартном цикле с применением настольной системы секвенирования MiniSeq™. Медленное время включения составляет от 40 до 50 секунд. В настоящей заявке быстрое время цикла относится к этапу включения, который составляет от 10 секунд до 40 секунд. В одном варианте реализации быстрое время цикла представляет собой время включения, составляющее не более 40 секунд, не более 30 секунд, не более 20 секунд, не более 18 секунд, не более 16 секунд, не более 14 секунд или не более 12 секунд. В одном варианте реализации быстрое время цикла представляет собой время включения, составляющее по меньшей мере 10 секунд, по меньшей мере 12 секунд, по меньшей мере 14 секунд, по меньшей мере 16 секунд, по меньшей мере 18 секунд, по меньшей мере 20 секунд или по меньшей мере 30 секунд. В одном варианте реализации быстрое время цикла представляет собой время включения, составляющее менее 40 секунд, менее 30 секунд, менее 20 секунд, менее 18 секунд, менее 16 секунд, менее 14 секунд, менее 12 секунд или менее 10 секунд.
[038] Измененную полимеразу, описанную в настоящей заявке, можно применять в реакциях SBS для прогонов различной длины. «Прогон» относится к количеству нуклеотидов, идентифицированных на матрице. Прогон обычно включает прогон на основе первого праймера (например, праймера read1), который считывает одну цепь матрицы, и прогон на основе второго праймера (например, праймера read2), который считывает комплементарную цепь матрицы. В одном варианте реализации количество нуклеотидов, идентифицированных с применением первого или второго праймера, может составлять от 10 до 150 нуклеотидов. В одном варианте реализации количество нуклеотидов, идентифицированных с применением первого праймера или второго праймера, может составлять не более 150 нуклеотидов, не более 130 нуклеотидов, не более 110 нуклеотидов, не более 90 нуклеотидов, не более 70 нуклеотидов, не более 50 нуклеотидов, не более 30 нуклеотидов или не более 20 нуклеотидов. В одном варианте реализации количество нуклеотидов, идентифицированных с применением первого праймера или второго праймера, может составлять по меньшей мере 10, по меньшей мере 20, по меньшей мере 30, по меньшей мере 50, по меньшей мере 70, по меньшей мере 90, по меньшей мере 110 или по меньшей мере 130 нуклеотидов.
[039] В некоторых вариантах реализации измененная полимераза основана на ДНК-полимеразе семейства типа В. Измененная полимераза может быть основана, например, на архейной ДНК-полимеразе семейства В, ДНК-полимеразе-а человека или фаговой полимеразе.
[040] Архейные ДНК-полимеразы семейства В хорошо известны в данной области техники, как это проиллюстрировано в раскрытии патента США №8283149. В некоторых вариантах реализации архейная ДНК-полимераза представляет собой ДНК-полимеразу из гипертермофильных архей и является термостабильной.
[041] В некоторых вариантах реализации архейная ДНК-полимераза семейства В происходит из такого рода, как, например, Thermococcus, Pyrococcus или Methanococcus. Члены рода Thermococcus хорошо известны в данной области техники и включают, но не ограничиваются ими, Т. 4557, Т. barophilus, Т. gammatolerans, Т. onnurineus, Т. sibiricus, Т. kodakarensis, Т. gorgonarius и Т. waiotapuensis. Представители рода Pyrococcus хорошо известны в данной области техники и включают, но не ограничиваются ими, P. NA2, P. abyssi, P. furiosus, P. horikoshii, P. yayanosii, P. endeavori, P. glycovorans и P. woesei. Члены рода Methanococcus хорошо известны в данной области техники и включают, но не ограничиваются ими, М aeolicus, М. maripaludis, М. vannielii, М voltae, М. thermolithotrophicus и М. jannaschii.
[042] В одном варианте реализации измененная полимераза основана на полимеразе Vent®, Deep Vent®, 9°N, Pfu, KOD или Pab. Vent® и Deep Vent® представляют собой коммерческие названия, используемые для ДНК-полимераз семейства В, выделенных из гипертермофильных архей Thermococcus litoraiis, Полимераза 9°N представляет собой полимеразу семейства В, выделенную из Thermococcus sp. Полимераза Pfu представляет собой полимеразу семейства В, выделенную из Pyrococcus furiosus. Полимераза KOD представляет собой полимеразу семейства В, выделенную из Thermococcus kodakaraenis. Полимераза Pab представляет собой полимеразу семейства В, выделенную из Pyrococcus abyssi. Twa представляет собой полимеразу семейства В, выделенную из Т. waiotapuensis. Примеры полимераз Vent®, Deep Vent®, 9°N, Pfu, KOD, Pab и Twa раскрыты на ФИГ. 1.
[043] В некоторых вариантах реализации архейная ДНК-полимераза семейства В происходит из фага, такого как, например, фаг Т4, RB69 или phi29.
[044] На ФИГ. 1 показано выравнивание последовательностей белков, имеющих аминокислотные последовательности, показанные в SEQ ID NO: 1-7. Выравнивание указывает на аминокислоты, которые являются консервативными в различных полимеразах семейства В. Специалисту в данной области техники будет понятно, что консервативные аминокислоты и консервативные области, скорее всего, являются консервативными, поскольку они важны для функционирования полимераз и, следовательно, демонстрируют корреляцию между структурой и функционированием полимераз. Выравнивание также показывает области вариабельности в различных полимеразах семейства В. Специалист в данной области техники может установить из таких данных области полимеразы, в которых замены, в частности консервативные замены, могут быть разрешены без чрезмерного воздействия на биологическую активность измененной полимеразы.
[045] Измененная полимераза, описанная в настоящей заявке, является полимеразой на основе аминокислотной последовательности известной полимеразы (также называемой в настоящей заявке эталонной полимеразой) и дополнительно включает мутации по типу замены в одном или более остатках. В одном варианте реализации мутация по типу замены находится в положении, функционально эквивалентном аминокислоте эталонной полимеразы. Под «функционально эквивалентным» подразумевается, что измененная полимераза имеет аминокислотную замену в аминокислотном положении в эталонной полимеразе, которая играет одинаковую функциональную роль как в эталонной полимеразе, так и в измененной полимеразе.
[046] В общем, функционально эквивалентные мутации по типу замены в двух или более различных полимеразах возникают в гомологичных аминокислотных положениях в аминокислотных последовательностях полимераз. Следовательно, использование в настоящей заявке термина «функционально эквивалентный» также охватывает мутации, которые являются «позиционно эквивалентными» или «гомологичными» данной мутации, независимо от того, известна ли конкретная функция мутированной аминокислоты. Можно идентифицировать местоположения функционально эквивалентных и позиционно эквивалентных аминокислотных остатков в аминокислотных последовательностях двух или более различных полимераз на основе выравнивания последовательностей и/или молекулярного моделирования. Пример выравнивания последовательностей для идентификации позиционно эквивалентных и/или функционально эквивалентных остатков представлен на ФИГ. 1. Например, остатки в полимеразах Twa, KOD, Pab, Pfu, Deep Vent и Vent на ФИГ. 1, которые выровнены по вертикали, считаются позиционно эквивалентными, а также функционально эквивалентными соответствующему остатку в аминокислотной последовательности полимеразы 9°N. Таким образом, например, остаток 349 в полимеразах 9°N, Twa, KOD, Pfu, Deep Vent и Pab и остаток 351 в полимеразе Vent являются функционально эквивалентными и позиционно эквивалентными. Подобным образом, например, остаток 633 в полимеразах 9°N, Twa, KOD и Pab, остаток 634 в полимеразах Pfu и Deep Vent и остаток 636 в полимеразе Vent являются функционально эквивалентными и позиционно эквивалентными. Специалист в данной области техники может легко идентифицировать функционально эквивалентные остатки в ДНК-полимеразах.
[047] В некоторых вариантах реализации мутация по типу замены включает мутацию до остатка, имеющего неполярную боковую цепь. Аминокислоты, имеющие неполярные боковые цепи, хорошо известны в данной области техники и включают, например: аланин, глицин, изолейцин, лейцин, метионин, фенилаланин, пролин, триптофан и валин.
[048] В некоторых вариантах реализации мутация по типу замены включает мутацию до остатка, имеющего полярную боковую цепь. Аминокислоты, имеющие полярные боковые цепи, хорошо известны в данной области техники и включают, например: аргинин, аспарагин, аспарагиновую кислоту, глутамин, глутаминовую кислоту, гистидин, лизин, серии, цистеин, тирозин и треонин.
[049] В некоторых вариантах реализации мутация по типу замены включает мутацию до остатка, имеющего гидрофобную боковую цепь. Аминокислоты, имеющие гидрофобные боковые цепи, хорошо известны в данной области техники и включают, например: глицин, аланин, валин, лейцин, изолейцин, пролин, фенилаланин, метионин и триптофан.
[050] В некоторых вариантах реализации мутация по типу замены включает мутацию до остатка, имеющего незаряженную боковую цепь. Аминокислоты с незаряженными боковыми цепями хорошо известны в данной области техники и включают, например: глицин, серии, цистеин, аспарагин, глутамин, тирозин и треонин, среди прочих.
[051] В одном варианте реализации измененная полимераза имеет аминокислотную последовательность, которая является сходной по структуре с эталонной полимеразой, раскрытой в настоящей заявке. В одном варианте реализации эталонная полимераза представляет собой полимеразу, которая включает аминокислотную последовательность 9°N (SEQ ID NO: 1). Необязательно, эталонная полимераза представляет собой SEQ ID NO: 1 со следующими мутациями по типу замены: Met129Ala, Asp141 Ala, Glu143Ala, Cys223Ser, Leu408Ala, Tyr409Ala, Pro410lle и Ala485Val. Полимераза, имеющая аминокислотную последовательность 9°N (SEQ ID NO: 1) с мутациями по типу замены Met129Ala, Asp141Ala, Glu143Ala, Cys223Ser, Leu408Ala, Tyr409Ala, Pro41Olle и Ala485Val, раскрыта в SEQ ID NO: 8 и также упоминается в настоящей заявке как полимераза Ро1812. Другие эталонные последовательности включают SEQ ID NO: 2, 3, 4, 5, 6 или 7. Необязательно, эталонная полимераза представляет собой SEQ ID NO: 2, 3, 4, 5, 6 или 7 с мутациями по типу замены, функционально и позиционно эквивалентными следующим мутациям по типу замены в SEQ ID NO: 1: Met129Ala, Asp141Ala, Glu143Ala, Cys223Ser, Leu408Ala, Tyr409Ala, Pro410lle и Ala485Val.
[052] В настоящей заявке измененная полимераза может быть «сходной по структуре» с эталонной полимеразой, если аминокислотная последовательность измененной полимеразы обладает определенной степенью сходства последовательностей и/или идентичности последовательностей по сравнению с эталонной полимеразой.
[053] Сходство по структуре двух аминокислотных последовательностей может быть определено путем выравнивания остатков двух последовательностей (например, полимеразы-кандидата и эталонной полимеразы, описанных в настоящей заявке) для оптимизации количества идентичных аминокислот по длине их последовательностей; при выравнивании допускаются пробелы в одной или обеих последовательностях, чтобы оптимизировать количество идентичных аминокислот, хотя аминокислоты в каждой последовательности, тем не менее, должны оставаться в своем правильном порядке. Полимераза-кандидат представляет собой полимеразу, которую сравнивают с эталонной полимеразой. Полимераза-кандидат, которая имеет сходство по структуре с эталонной полимеразой и полимеразную активность, представляет собой измененную полимеразу.
[054] Если в настоящей заявке не указано иное, анализ попарного сравнения аминокислотных последовательностей или нуклеотидных последовательностей может быть проведен, например, с помощью алгоритма локальной гомологии Smith & Waterman, Adv. Appl. Math. 2:482 (1981), by the homology alignment algorithm of Needleman & Wunsch, J. Mol. Biol. 48:443 (1970), способом поиска сходства Pearson & Lipman, Proc. Nat'l. Acad. Sci. USA 85:2444 (1988), с помощью компьютеризированной реализации данных алгоритмов (GAP, BESTFIT, FASTA и TFASTA в пакете программного обеспечения Wisconsin Genetics Software Package, Genetics Computer Group, 575 Science Dr., Madison, Wis.) или с помощью визуальной оценки (см. в целом, Current Protocols in Molecular Biology, Ausubel et al., eds., Current Protocols, a joint venture between Greene Publishing Associates, Inc. and John Wiley & Sons, Inc., дополненное в 2004).
[055] Одним из примеров алгоритма, который подходит для определения сходства по структуре, является алгоритм BLAST, который описан в Altschul et al., J. Mol. Biol. 215:403-410 (1990). Программное обеспечение для проведения анализов BLAST находится в общем доступе через Национальный центр биотехнологической информации. Данный алгоритм включает в себя сначала идентификацию пар последовательностей с высоким показателем сходства (HSP) путем идентификации коротких «слов» длины W в запрашиваемой последовательности, которые либо совпадают, либо удовлетворяют некоторой положительной пороговой оценке Т при выравнивании со «словом» такой же длины в последовательности из базы данных. Т называется порогом показателя сходства соседних слов (Altschul et al., J. Mol. Biol. 215:403-410 (1990)). Эти первоначальные совпадения соседних «слов» играют роль затравок для инициирования поисков с целью обнаружения более длинных HSP, содержащих их. Затем совпадения слов продлевают в обоих направлениях вдоль каждой последовательности до тех пор, пока суммарный балл выравнивания может увеличиваться. Суммарные баллы вычисляют, используя для нуклеотидных последовательностей параметры М (призовой балл за пару совпадающих остатков; всегда больше 0) и N (штрафной балл за несовпадающие остатки; всегда меньше 0). Для аминокислотных последовательностей матрицу подсчета баллов применяют для вычисления суммарного балла. Совпадения слов прекращают продлевать в каждом направлении, если: суммарный балл выравнивания снижается на величину X от максимальной достигнутой им величины; суммарный балл опускается до нуля или ниже, вследствие накопления одного или более выравниваний остатков с отрицательными баллами или достигается конец любой последовательности. Параметры W, Т и X алгоритма BLAST определяют чувствительность и скорость выравнивания. В программе BLASTN (для нуклеотидных последовательностей) в качестве параметров по умолчанию используются длина слова (W), равная 11, ожидание (Е), равное 10, отсечка, равная 100, М равное 5, N равное -4 и сравнение обеих нитей. Для аминокислотных последовательностей в программе BLASTP в качестве параметров по умолчанию используются длина слова (W), равная 3, ожидание (Е), равное 10, и матрица подсчета баллов BLOSUM62 (см. Henikoff & Henikoff (1989) Proc. Natl. Acad. Sci. USA 89:10915).
[056] В дополнение к вычислению процентной идентичности последовательностей алгоритм BLAST также выполняет статистический анализ сходства между двумя последовательностями (см., например, Karlin & Altschul, Proc. Nat'l. Acad. Sci. USA 90:5873- 5787 (1993)). Один критерий сходства, предоставляемый алгоритмом BLAST, представляет собой вероятность наименьшей суммы (P(N)), который является показателем вероятности, с которой совпадение между двумя нуклеотидными или аминокислотными последовательностями произошло бы случайно. Например, нуклеиновую кислоту считают сходной с эталонной последовательностью, если вероятность наименьшей суммы при сравнении тестируемой нуклеиновой кислоты с эталонной нуклеиновой кислотой составляет менее примерно 0,1, более предпочтительно менее примерно 0,01 и наиболее предпочтительно менее примерно 0,001.
[057] При сравнении двух аминокислотных последовательностей сходство по структуре может быть обозначено процентом «идентичности» или может быть обозначено процентом «сходства». «Идентичность» относится к наличию идентичных аминокислот.«Сходство» относится к наличию не только идентичных аминокислот, но также к наличию консервативных замен. Консервативная замена аминокислоты в белке может быть выбрана из других членов класса, к которому принадлежит аминокислота. Например, в области биохимии белков хорошо известно, что аминокислота, принадлежащая к группе аминокислот, имеющих определенный размер или характеристики (такие как заряд, гидрофобность или гидрофильность), может быть заменена другой аминокислотой без изменения активности белка, особенно в областях белка, которые напрямую не связаны с биологической активностью. Например, неполярные аминокислоты включают аланин, глицин, изолейцин, лейцин, метионин, фенилаланин, пролин, триптофан и валин. Гидрофобные аминокислоты включают глицин, аланин, валин, лейцин, изолейцин, пролин, фенилаланин, метионин и триптофан. Полярные аминокислоты включают аргинин, аспарагин, аспарагиновую кислоту, глутамин, глутаминовую кислоту, гистидин, лизин, серии, цистеин, тирозин и треонин. Незаряженные аминокислоты включают, среди прочего, глицин, серии, цистеин, аспарагин, глутамин, тирозин и треонин.
[058] Таким образом, в настоящей заявке ссылка на полимеразу, описанную в настоящей заявке, такая как ссылка на аминокислотную последовательность, представляющую собой одну или более SEQ ID NO, описанных в настоящей заявке, может включать в себя белок со сходством последовательностей аминокислот с эталонной полимеразой, составляющим по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 86%, по меньшей мере 87%, по меньшей мере 88%, по меньшей мере 89%, по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99%.
[059] В качестве альтернативы, в настоящей заявке ссылка на полимеразу, описанную в настоящей заявке, такая как ссылка на аминокислотную последовательность, представляющую собой одну или более SEQ ID NO, описанных в настоящей заявке, может включать в себя белок с идентичностью последовательностей аминокислот с эталонной полимеразой, составляющей по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 86%, по меньшей мере 87%, по меньшей мере 88%, по меньшей мере 89%, по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99%.
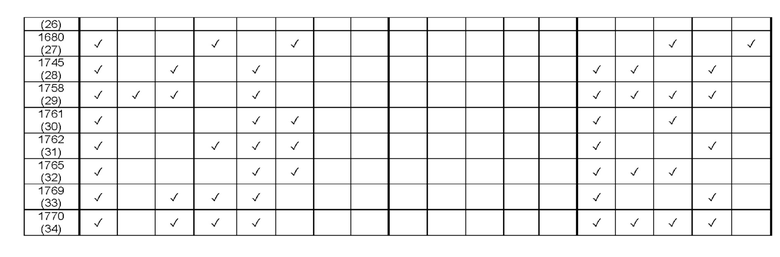
[060] В настоящем раскрытии описан набор мутаций, которые приводят к получению полимеразы, имеющей одну или более из описанных в настоящей заявке активностей. Полимераза, описанная в настоящей заявке, может включать любое количество мутаций, например, по меньшей мере 1, по меньшей мере 2, по меньшей мере 3, по меньшей мере 4, по меньшей мере 5, по меньшей мере 6, по меньшей мере 7, по меньшей мере 8, по меньшей мере 9, по меньшей мере по меньшей мере 10, по меньшей мере 11, по меньшей мере 12, по меньшей мере 13, по меньшей мере 14, по меньшей мере 15, по меньшей мере 16, по меньшей мере 17 или по меньшей мере 18 мутаций по сравнению с эталонной полимеразой, такой как SEQ ID NO: 1 или SEQ ID NO: 8. Аналогичным образом, описанная в настоящей заявке полимераза может включать мутации в любой комбинации. Например, в таблице 1 приведены примеры конкретных измененных полимераз, которые включают различные комбинации описанных в настоящей заявке мутаций. Галочка (√) обозначает наличие указанной мутации. Указанные мутации, например, Y497G, F152G, V278L и т.д., представляют собой мутации в положениях в SEQ ID NO: 1.
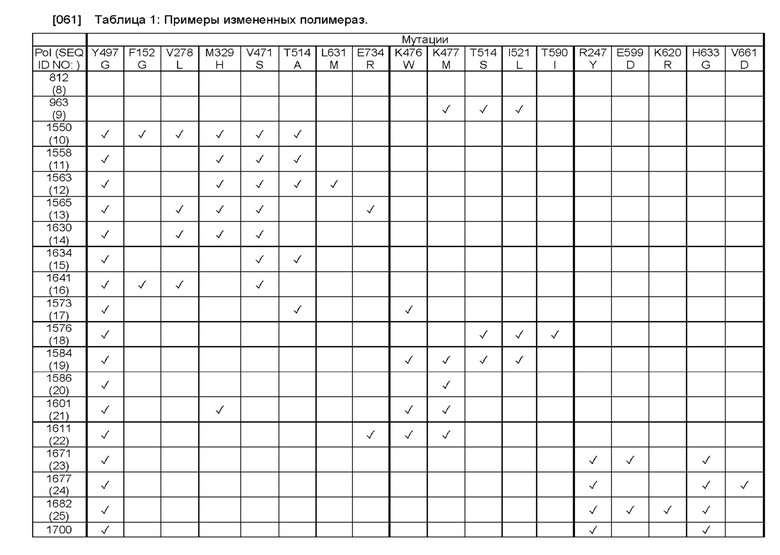
[062] Измененная полимераза согласно настоящему раскрытию включает мутацию по типу замены в положении, функционально эквивалентном Туг497 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Туг497, представляет собой мутацию до неполярной, гидрофобной или незаряженной аминокислоты, например, Gly.
[063] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Рпе152 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Phe152, представляет собой мутацию до неполярной, гидрофобной или незаряженной аминокислоты, например, Gly.
[064] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Val278 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Val278, представляет собой мутацию до неполярной или гидрофобной аминокислоты, например, Leu.
[065] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Met329 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Met329, представляет собой мутацию до полярной аминокислоты, например, His.
[066] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Val471 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Val471, представляет собой мутацию до полярной или незаряженной аминокислоты, например, Ser.
[067] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Thr514 в полимеразе 9°N (SEQ ID NO: 1), как известно в данной области техники и проиллюстрировано в заявке на патент США №2016/0032377. В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Thr514, представляет собой мутацию до неполярной или гидрофобной аминокислоты, например, Ala. В некоторых вариантах реализации другие мутации по типу замены, которые можно применять в комбинации с неполярной или гидрофобной аминокислотой в положении, функционально эквивалентном Thr514, включают Phe152, Val278, М329, Val471, Lue631, Glu734 или их комбинацию. В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Thr514, представляет собой мутацию до полярной или незаряженной аминокислоты, например, Ser. В некоторых вариантах реализации другие мутации по типу замены, которые можно применять в комбинации с полярной или незаряженной аминокислотой в положении, функционально эквивалентном Thr514, включают Lys476, Lys477, lle521, Thr590 или их комбинацию.
[068] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Leu631 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Leu631, представляет собой мутацию до неполярной или гидрофобной аминокислоты, например, Met.
[069] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Glu734 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Glu734, представляет собой мутацию до полярной или незаряженной аминокислоты, например, Arg.
[070] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Lys476 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Lys476, представляет собой мутацию до неполярной или гидрофобной аминокислоты, например, Trp.
[071] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Lys477 в полимеразе 9°N (SEQ ID NO: 1), как известно в данной области техники и проиллюстрировано в раскрытии патента США №9765309. В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Lys477, представляет собой мутацию до неполярной или гидрофобной аминокислоты, например, Met.
[072] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Ие521 в полимеразе 9°N (SEQ ID NO: 1), как известно в данной области техники и проиллюстрировано в заявке на патент США №2016/0032377. В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Пе521, представляет собой мутацию до неполярной аминокислоты, например, Leu.
[073] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Thr590 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Thr590, представляет собой мутацию до неполярной или гидрофобной аминокислоты, например, lle.
[074] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Arg247 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Arg247, представляет собой мутацию до неполярной или незаряженной аминокислоты, например, Tyr.
[075] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Glu599 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Glu599, представляет собой мутацию до полярной или незаряженной аминокислоты, например, Asp.
[076] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Lys620 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Lys620, представляет собой мутацию до полярной или незаряженной аминокислоты, например, Arg.
[077] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном His633 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном His633, представляет собой мутацию до неполярной, гидрофобной или незаряженной аминокислоты, например, Gly.
[078] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Val661 в полимеразе 9°N (SEQ ID NO: 1). В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Val661, представляет собой мутацию до полярной или незаряженной аминокислоты, например, Asp.
[079] В одном варианте реализации измененная полимераза включает по меньшей мере одну мутацию по типу замены в положении, функционально эквивалентном аминокислотам Met129, Asp141, Glu143, Cys223, Lys408, Tyr409, Pro410, Ala485, или их комбинацию. В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Met129, Asp141, Glu143, Lys408 или Tyr409, представляет собой мутацию до неполярной или гидрофобной аминокислоты, например, Ala. В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Cys223, представляет собой мутацию до полярной или незаряженной аминокислоты, например, Ser. В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном Pro410, представляет собой мутацию до неполярной или гидрофобной аминокислоты, например, lle. В одном варианте реализации мутация по типу замены в положении, функционально эквивалентном А1а485, представляет собой мутацию до неполярной или гидрофобной аминокислоты, например, Val.
[080] В одном варианте реализации измененная полимераза включает мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, и по меньшей мере одну, по меньшей мере две, по меньшей мере три, по меньшей мере четыре, по меньшей мере пять, по меньшей мере шесть или семь мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Phe152, Val278, Met329, Val471, Thr514, Leu631 и Glu734 в аминокислотной последовательности ДНК-полимеразы 9°N. В одном варианте реализации измененная полимераза также включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[081] В одном варианте реализации измененная полимераза включает мутацию по типу замены аминокислоты в положении, функционально эквивалентном Туг497, и по меньшей мере одну, по меньшей мере две, по меньшей мере три, по меньшей мере четыре или пять мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Lys476, Lys477, Thr514, lle521 и Thr590 в аминокислотной последовательности ДНК-полимеразы 9°N. В одном варианте реализации измененная полимераза также включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[082] В одном варианте реализации измененная полимераза включает мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, и по меньшей мере две, по меньшей мере три, по меньшей мере четыре или пять мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Arg247, Glu599, Lys620, His633 и Val661 в аминокислотной последовательности ДНК-полимеразы 9°N. В одном варианте реализации измененная полимераза также включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[083] В одном варианте реализации измененная полимераза включает мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, по меньшей мере одну, по меньшей мере две, по меньшей мере три, по меньшей мере четыре, по меньшей мере пять, по меньшей мере шесть или семь мутаций по типу замены аминокислоты в положении, функционально эквивалентном Phe152, Val278, Met329, Val471, Thr514, Leu631 или Glu734 в аминокислотной последовательности ДНК-полимеразы 9°N, и (iii) по меньшей мере одну, по меньшей мере две, по меньшей мере три, по меньшей мере четыре или пять мутаций по типу замены аминокислоты в положении, функционально эквивалентном Lys476, Lys477, Thr514, Ile521 или Thr590 в аминокислотной последовательности ДНК-полимеразы 9°N. В одном варианте реализации измененная полимераза также включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[084] В одном варианте реализации измененная полимераза включает мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, по меньшей мере одну, по меньшей мере две, по меньшей мере три, по меньшей мере четыре, по меньшей мере пять, по меньшей мере шесть или семь мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Phe152, Val278, Met329, Val471, Thr514, Leu631 и Glu734 в аминокислотной последовательности ДНК-полимеразы 9°N, и по меньшей мере одну, по меньшей мере две, по меньшей мере три, по меньшей мере четыре или пять мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Arg247, Glu599, Lys620, His633 или Val661 в аминокислотной последовательности ДНК-полимеразы 9°N. В одном варианте реализации измененная полимераза также включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[085] В одном варианте реализации измененная полимераза включает мутацию по типу замены аминокислоты в положении, функционально эквивалентном Туг497, по меньшей мере одну, по меньшей мере две, по меньшей мере три, по меньшей мере четыре или пять мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Lys476, Lys477, Thr514, Ile521 или Thr590 в аминокислотной последовательности ДНК-полимеразы 9°N, и по меньшей мере одну, по меньшей мере две, по меньшей мере три, по меньшей мере четыре или пять мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Arg247, Glu599, Lys620, His633 или Val661 в аминокислотной последовательности ДНК-полимеразы 9°N. В одном варианте реализации измененная полимераза также включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[086] В одном варианте реализации измененная полимераза включает мутацию по типу замены аминокислоты в положении, функционально эквивалентном Туг497, по меньшей мере одну, по меньшей мере две, по меньшей мере три, по меньшей мере четыре, по меньшей мере пять или шесть мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Phe152, Val278, Met329, Val471, Leu631 и Glu734 в аминокислотной последовательности ДНК-полимеразы 9°N, по меньшей мере одну, по меньшей мере две, по меньшей мере три, по меньшей мере четыре или пять мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Lys476, Lys477, Thr514, Ile521 или Thr590 в аминокислотной последовательности ДНК-полимеразы 9°N, и по меньшей мере одну, по меньшей мере две, по меньшей мере три, по меньшей мере четыре или пять мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Arg247, Glu599, Lys620, His633 или Val661 в аминокислотной последовательности ДНК-полимеразы 9°N. В одном варианте реализации измененная полимераза также включает мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
[087] Конкретные примеры измененных полимераз включают Pol 1550 (SEQ ID NO: 10), Pol 1558 (SEQ ID NO: 11), Pol 1563 (SEQ ID NO: 12), Pol 1565 (SEQ ID NO: 13), Pol 1630 (SEQ ID NO: 14), Pol 1634 (SEQ ID NO: 15), Pol 1641 (SEQ ID NO: 16), Pol 1573 (SEQ ID NO: 17), Pol 1576 (SEQ ID NO: 18), Pol 1584 (SEQ ID NO: 19), Pol 1586 (SEQ ID NO: 20), Pol 1601 (SEQ ID NO: 21), Pol 1611 (SEQ ID NO: 22), Pol 1671 (SEQ ID NO: 23), Pol 1677 (SEQ ID NO: 24), Pol 1682 (SEQ ID NO: 25), Pol 1680 (SEQ ID NO: 27), Pol 1745 (SEQ ID NO: 28), Pol 1758 (SEQ ID NO: 29), Pol 1761 (SEQ ID NO: 30), Pol 1762 (SEQ ID NO: 31), Pol 1765 (SEQ ID NO: 32), Pol 1769 (SEQ ID NO: 33) и Pol 1770 (SEQ ID NO: 34).
[088] Измененная полимераза, описанная в настоящей заявке, может включать дополнительные мутации, которые, как известно, влияют на активность полимеразы. При такой замене мутация находится в положении, функционально эквивалентном Arg713 в полимеразе 9°N (SEQ ID NO: 1). Может быть осуществлена любая из множества мутаций по типу замены в одном или более положениях, которые, как известно, приводят к снижению экзонуклеазной активности, как это известно в данной области техники и проиллюстрировано патентом США №8623628. В одном варианте реализации мутация по типу замены в положении Arg713 представляет собой мутацию до неполярной, гидрофобной или незаряженной аминокислоты, например, Gly, Met или Ala.
[089] В одном варианте реализации измененная полимераза включает мутацию по типу замены в положении, функционально эквивалентном Arg743 или Lys705, или их комбинацию в полимеразе 9°N (SEQ ID NO: 1), как известно в данной области техники и проиллюстрировано в раскрытии патента США №8623628. В одном варианте реализации мутация по типу замены в положении Arg743 или Lys705 представляет собой мутацию до неполярной или гидрофобной аминокислоты, например, Ala.
[090] В настоящем раскрытии также предложены композиции, которые включают измененную полимеразу, описанную в настоящей заявке. Композиция может включать другие компоненты, помимо измененной полимеразы. Например, композиция может включать буфер, раствор нуклеотидов или их комбинацию. Раствор нуклеотидов может включать нуклеотиды, такие как меченые, синтетические, модифицированные нуклеотиды или их комбинацию. В одном варианте реализации композиция включает целевые нуклеиновые кислоты, такие как библиотека целевых нуклеиновых кислот.
Мутирующие полимеразы
[091] В настоящем раскрытии необязательно применяют различные типы мутагенеза, например, для модификации полимераз для получения вариантов, например, в соответствии с моделями полимеразы и прогнозированием на основе моделей, как описано выше, или с применением подходов случайных или полуслучайных мутаций. В целом, для получения мутантов полимеразы можно применять любую доступную процедуру мутагенеза. Такие процедуры мутагенеза необязательно включают отбор мутантных нуклеиновых кислот и полипептидов для одной или более представляющих интерес активностей (например, сниженного пирофосфолиза, повышенного оборота, например, для данного аналога нуклеотида). Процедуры, которые можно применять, включают, но не ограничиваются ими: сайт-направленный точечный мутагенез, случайный точечный мутагенез, гомологичную рекомбинацию in vitro или in vivo (перестановка в ДНК и комбинированная ПЦР с перекрывающимися праймерами), мутагенез с применением урацилсодержащих матриц, олигонуклеотид-направленный мутагенез, фосфоротиоат-модифицированный мутагенез ДНК, мутагенез с применением дуплексной ДНК с пробелом, репарацию точечного несоответствия, мутагенез с применением штаммов-хозяев с дефицитом репарации, рестрикцию-селекцию и рестрикцию-очистку, делеционный мутагенез, мутагенез посредством синтеза полного гена, вырожденную ПЦР, репарацию двухцепочечных разрывов, и многие другие, известные специалистам в данной области техники. Исходной полимеразой для мутации может быть любая из указанных в настоящей заявке, включая доступные мутанты полимеразы, такие как идентифицированные, например, в патенте США №8460910 и патенте США №8623628, каждый из которых во всей полноте включен в настоящую заявку посредством ссылки.
[092] Необязательно, мутагенез можно проводить с применением известной информации о встречающейся в природе молекуле полимеразы или известной измененной или мутированной полимеразе (например, с применением существующей мутантной полимеразы), например, последовательности, сравнении последовательностей, физических свойствах, кристаллической структуре и/или как обсуждалось выше. Однако в другом классе вариантов реализации модификация может быть по существу случайной (например, как в классической или "family" перестановке ДНК, см., например, Crameri et al. (1998) "DNA shuffling of a family of genes from diverse species accelerates directed evolution" Nature 391:288-291).
[093] Дополнительную информацию о форматах мутаций можно найти в: Sambrook et al., Molecular Cloning-A Laboratory Manual (3rd Ed.), Vol.1-3, Cold Spring Harbor Laboratory, Cold Spring Harbor, N.Y., 2000 ("Sambrook"); Current Protocols in Molecular Biology, F. M. Ausubel et al., eds., Current Protocols, a joint venture between Greene Publishing Associates, Inc. and John Wiley & Sons, Inc., (дополнено в 2011) ("Ausubel")) и PCR Protocols A Guide to Methods and Applications (Innis et al. eds) Academic Press Inc. San Diego, Calif. (1990) ("Innis"). Следующие процитированные публикации и ссылки предоставляют дополнительные подробности о форматах мутаций: Arnold, Protein engineering for unusual environments, Current Opinion in Biotechnology 4:450-455 (1993); Bass et al., Mutant Trp repressors with new DNA-binding specificities, Science 242:240-245 (1988); Bordo and Argos (1991) Suggestions for "Safe" Residue Substitutions in Site-directed Mutagenesis 217:721-729; Botstein & Shortle, Strategies and applications of in vitro mutagenesis, Science 229:1193-1201 (1985); Carter et al., Improved oligonucleotide site-directed mutagenesis using M13 vectors, Nucl. Acids Res. 13: 4431 -4443 (1985); Carter, Site-directed mutagenesis, Biochem. J. 237:1-7 (1986); Carter, Improved oligonucleotide-directed mutagenesis using M13 vectors, Methods in Enzymol. 154: 382-403 (1987); Dale et al., Oligonucleotide-directed random mutagenesis using the phosphorothioate method, Methods Mol. Biol. 57:369-374 (1996); Eghtedarzadeh & Henikoff, Use of oligonucleotides to generate large deletions, Nucl. Acids Res. 14: 5115 (1986); Fritz et al., Oligonucleotide-directed construction of mutations: a gapped duplex DNA procedure without enzymatic reactions in vitro, Nucl. Acids Res. 16: 6987-6999 (1988); Grundstrom et al., Oligonucleotide-directed mutagenesis by microscale 'shot-gun' gene synthesis, Nucl. Acids Res. 13: 3305-3316 (1985); Hayes (2002) Combining Computational and Experimental Screening for rapid Optimization of Protein Properties PNAS 99(25) 15926-15931; Kunkel, The efficiency of oligonucleotide directed mutagenesis, in Nucleic Acids & Molecular Biology (Eckstein, F. and Lilley, D. M. J. eds., Springer Verlag, Berlin)) (1987); Kunkel, Rapid and efficient site-specific mutagenesis without phenotypic selection, Proc. Natl. Acad. Sci. USA 82:488-492 (1985); Kunkel et al. Rapid and efficient site-specific mutagenesis without phenotypic selection, Methods in Enzymol. 154, 367-382 (1987); Kramer et al. The gapped duplex DNA approach to oligonucleotide-directed mutation construction, Nucl. Acids Res. 12: 9441-9456 (1984); Kramer & Fritz Oligonucleotide-directed construction of mutations via gapped duplex DNA, Methods in Enzymol. 154:350-367 (1987); Kramer et al. Point Mismatch Repair, Cell 38:879-887 (1984); Kramer et al. Improved enzymatic in vitro reactions in the gapped duplex DNA approach to oligonucleotide-directed construction of mutations, Nucl. Acids Res. 16: 7207 (1988); Ling et al. Approaches to DNA mutagenesis: an overview, Anal Biochem. 254(2): 157-178 (1997); Lorimer and Pastan Nucleic Acids Res. 23, 3067-8 (1995); Mandecki, Oligonucleotide-directed double-strand break repair in plasmids of Escherichia coli: a method for site-specific mutagenesis, Proc. Natl. Acad. Sci. USA, 83:7177-7181(1986); Nakamaye & Eckstein, Inhibition of restriction endonuclease Nci I cleavage by phosphorothioate groups and its application to oligonucleotide-directed mutagenesis, Nucl. Acids Res. 14: 9679-9698 (1986); Nambiar et al. Total synthesis and cloning of a gene coding for the ribonuclease S protein, Science 223: 1299-1301(1984); Sakamar and Khorana, Total synthesis and expression of a gene for the a-subunit of bovine rod outer segment guanine nucleotide-binding protein (transducin), Nucl. Acids Res. 14: 6361-6372 (1988); Sayers et al, Y-T Exonucleases in phosphorothioate-based oligonucleotide-directed mutagenesis, Nucl. Acids Res. 16:791-802 (1988); Sayers et al. Strand specific cleavage of phosphorothioate-containing DNA by reaction with restriction endonucleases in the presence of ethidium bromide, (1988) Nucl. Acids Res. 16: 803-814; Sieber, et al. Nature Biotechnology, 19:456-460 (2001); Smith, In vitro mutagenesis, Ann. Rev. Genet. 19:423-462 (1985); Methods in Enzymol. 100: 468-500 (1983); Methods in Enzymol. 154: 329-350 (1987); Stemmer, Nature 370, 389-91(1994); Taylor et al. The use of phosphorothioate-modified DNA in restriction enzyme reactions to prepare nicked DNA, Nucl. Acids Res. 13: 8749-8764 (1985); Taylor et al. The rapid generation of oligonucleotide-directed mutations at high frequency using phosphorothioate-modified DNA, Nucl. Acids Res. 13: 8765-8787 (1985); Wells et al. Importance of hydrogen-bond formation in stabilizing the transition state of subtilisin, Phil. Trans. R. Soc. Lond. A 317: 415-423 (1986); Wells et al. Cassette mutagenesis: an efficient method for generation of multiple mutations at defined sites, Gene 34:315-323 (1985); Zoller & Smith, Oligonucleotide-directed mutagenesis using M 13-derived vectors: an efficient and general procedure for the production of point mutations in any DNA fragment, Nucleic Acids Res. 10:6487-6500 (1982); Zoller & Smith, Oligonucleotide-directed mutagenesis of DNA fragments cloned into M13 vectors, Methods in Enzymol. 100:468-500 (1983); Zoller & Smith, Oligonucleotide-directed mutagenesis: a simple method using two oligonucleotide primers and a single-stranded DNA template, Methods in Enzymol. 154:329-350 (1987); Clackson et al. (1991) "Making antibody fragments using phage display libraries" Nature 352:624-628; Gibbs et al. (2001) "Degenerate oligonucleotide gene shuffling (DOGS): a method for enhancing the frequency of recombination with family shuffling" Gene 271:13-20; и Hiraga and Arnold (2003) "General method for sequence-independent site-directed chimeragenesis: J. Mol. Biol. 330:287-296. Дополнительные сведения о многих из вышеперечисленных способов можно найти в Methods in Enzymology Volume 154, где также описаны подходящие варианты контроля для устранения проблем с помощью различных способов мутагенеза.
Получение и выделение рекомбинантных полимераз
[094] В целом, нуклеиновые кислоты, кодирующие полимеразу, представленную в настоящей заявке, могут быть получены путем клонирования, рекомбинации, синтеза in vitro, амплификации in vitro и/или других доступных способов. Для экспрессии вектора экспрессии, который кодирует полимеразу, представленную в настоящей заявке, можно применять различные рекомбинантные способы. Способы получения рекомбинантных нуклеиновых кислот, экспрессии и выделения экспрессируемых продуктов хорошо известны и описаны в данной области техники. В настоящей заявке описан ряд примеров мутаций и комбинаций мутаций, а также стратегии создания желаемых мутаций. Способы создания и отбора мутаций в активном сайте полимераз, в том числе для модификации стерических свойств в активном сайте или рядом с ним, чтобы обеспечить улучшенный доступ для аналогов нуклеотидов, можно найти в настоящей заявке и, например, в WO 2007/076057 и WO 2008/051530.
[095] Дополнительные полезные ссылки на способы мутации, рекомбинантные способы и манипуляции с нуклеиновыми кислотами in vitro (включая клонирование, экспрессию, ПЦР и тому подобные) включают Berger and Kimmel, Guide to Molecular Cloning Techniques, Methods in Enzymology volume 152 Academic Press, Inc., San Diego, Calif. (Berger); Kaufman et al. (2003) Handbook of Molecular and Cellular Methods in Biology and Medicine Second Edition Ceske (ed) CRC Press (Kaufman); The Nucleic Acid Protocols Handbook Ralph Rapley (ed) (2000) Cold Spring Harbor, Humana Press Inc (Rapley); Chen et al. (ed) PCR Cloning Protocols, Second Edition (Methods in Molecular Biology, volume 192) Humana Press; и Viljoen et al. (2005) Molecular Diagnostic PCR Handbook Springer, ISBN 1402034032.
[096] Кроме того, множество наборов для очистки плазмид или других соответствующих нуклеиновых кислот от клеток являются коммерчески доступными (см, например, EasyPrep™ и FlexiPrep™, оба от Pharmacia Biotech; StrataClean™ от Stratagene; и QIAprep™ от Qiagen). Любая выделенная и/или очищенная нуклеиновая кислота может быть подвергнута дальнейшим манипуляциям для получения других нуклеиновых кислот, применяемых для трансфекции клеток, включения в родственные векторы для инфицирования организмов с целью экспрессии и/или тому подобного. Типичные векторы клонирования содержат терминаторы транскрипции и трансляции, последовательности инициации транскрипции и трансляции и промоторы, подходящие для регуляции экспрессии конкретной целевой нуклеиновой кислоты. Векторы необязательно содержат стандартные кассеты экспрессии, содержащие по меньшей мере одну независимую терминаторную последовательность, последовательности, обеспечивающие репликацию кассеты в эукариотах или прокариотах, или и то, и другое (например, челночные векторы) и селективные маркеры как для прокариотической, так и для эукариотической систем. Векторы подходят для репликации и интеграции в прокариотах, эукариотах или в обоих.
[097] Другие полезные ссылки, например для выделения и культивирования (например, для последующего выделения нуклеиновой кислоты) включают Freshney Culture of Animal Cells, a Manual of Basic Technique, third edition, Wiley-Liss, New ссылки, приведенные в ней; Payne et al. (1992) Plant Cell and Tissue Culture in Liquid S John Wiley & Sons, Inc. New York, N.Y.; Gamborg and Phillips (eds) (1995) Plant Cell, Tiss Organ Culture; Fundamental Methods Springer Lab Manual, Springer-Verlag (Berlin Hei New York); и Atlas and Parks (eds) The Handbook of Microbiological Media (1993) CRC Boca Raton, Fla.
[098] Настоящее раскрытие также включает нуклеиновые кислоты, кодирующие измененные полимеразы, описанные в настоящей заявке. Конкретная аминокислота быть закодирована множеством кодонов, и определенные системы трансляции (например, прокариотические или эукариотические клетки) часто демонстрируют предпочтение кс например, разные организмы часто предпочитают один из нескольких синонимичных кодонов, которые кодируют одну и ту же аминокислоту. По существу, нуклеиновые кислоты, представленные в настоящей заявке, необязательно являются «кодон-оптимизированными”, что означает, что нуклеиновые кислоты синтезируются с включением кодонов, которые являются предпочтительными для конкретной системы трансляции, применяемой для экспрессии полимеразы. Например, когда необходимо экспрессировать полимеразу в бактериальной клетке (или даже в конкретном штамме бактерий), нуклеиновая кислота может быть синтезирована с включением кодонов, наиболее часто встречающихся в геноме этой бактериальной клетки, для эффективной экспрессии полимеразы. Подобную стр можно применять, когда необходимо экспрессировать полимеразу в эукариотической клетке, например, нуклеиновая кислота может включать кодоны, предпочтительные для этой эукариотической клетки.
[099] Известны различные способы выделения и детектирования белков, которые можно применять для выделения полимераз, например, из рекомбинантных культур клеток, экспрессирующих рекомбинантные полимеразы, представленные в настоящей заявке. В данной области техники хорошо известны различные способы выделения и детектирования белков, в том числе, например, изложенные в R. Scopes, Protein Purification, Springer-N.Y. (1982); Deutscher, Methods in Enzymology Vol.182: Guide to Protein Purification, Ac Press, Inc. N.Y. (1990); Sandana (1997) Bioseparation of Proteins, Academic Press, Inc.; & al. (1996) Protein Methods, 2nd Edition Wiley-Liss, NY; Walker (1996) The Protein Protocols Handbook Humana Press, NJ, Harris and Angal (1990) Protein Purification Applications: A Practical Approach IRL Press at Oxford, Oxford, England; Harris and Angal Protein Purification Methods: A Practical Approach IRL Press at Oxford, Oxford, England; Scopes (1993) Protein Purification: Principles and Practice 3rd Edition Springer Verlag, NY; Janson and Ryden (1998) Protein Purification: Principles, High Resolution Methods and Applications, Second Edition Wiley-VCH, NY; и Walker (1998) Protein Protocols on CD-ROM Humana Press, NJ; и ссылках, приведенных в них. Дополнительные сведения о способах очистки и детектирования белков можно найти в издании Satinder Ahuja ed. Handbook of Bioseparations, Academic Press (2000).
Способы применения
[0100] Представленные в настоящей заявке измененные полимеразы можно применять в процедуре секвенирования, такой как способ секвенирования путем синтеза (SBS). Вкратце, SBS можно инициировать путем приведения целевых нуклеиновых кислот в контакт с одним или более нуклеотидами (например, мечеными, синтетическими, модифицированными или их комбинацией), ДНК-полимеразой и т.д. Те признаки, когда праймер удлиняется с применением целевой нуклеиновой кислоты в качестве матрицы, будут включать меченый нуклеотид, который можно детектировать. Время включения, применяемое в прогоне секвенирования, может быть значительно снижено с применением измененных полимераз, описанных в настоящей заявке. Необязательно, меченые нуклеотиды могут дополнительно включать свойство обратимой терминации, которое прекращает дальнейшее удлинение праймера после того, как нуклеотид был добавлен к праймеру. Например, к праймеру может быть добавлен аналог нуклеотида, имеющий обратимый терминаторный фрагмент, благодаря которому последующее удлинение не может происходить до тех пор, пока не будет доставлен агент для снятия блокировки для удаления фрагмента. Таким образом, для вариантов реализации, в которых применяют обратимую терминацию, реагент для снятия блокировки может быть доставлен в проточную ячейку (до или после детектирования). Промывку можно проводить между различными этапами доставки. Затем цикл можно повторить n раз для удлинения праймера на n нуклеотидов, тем самым проводя детектирование последовательности длиной n. Примеры процедур SBS, жидкостные системы и платформы детектирования, которые могут быть легко адаптированы для применения с чипом, полученным способами согласно настоящему раскрытию, описаны, например, в Bentley et al. Nature 456:53-59 (2008); WO 04/018497; WO 91/06678; WO 07/123744; в патентах США №7057026, 7329492, 7211414, 7315019, 7405281 и 8343746.
[0101] Можно применять другие процедуры секвенирования, в которых применяют циклические реакции, такие как пиросеквенирование. При пиросеквенировании детектируют высвобождение неорганического пирофосфата (PPi, от англ. inorganic pyrophosphate) по мере того, как определенные нуклеотиды включаются в формирующуюся цепь нуклеиновой кислоты (Ronaghi, et al. Analytical Biochemistry 242(1), 84-9 (1996); Ronaghi, Genome Res. 11(1), 3-11 (2001); Ronaghi et al. Science 281(5375), 363 (1998); патенты США №6210891; 6258568 и 6274320). При пиросеквенировании высвободившийся PPi можно детектировать путем превращения в аденозинтрифосфат (АТФ) с помощью АТФ-сульфурилазы, а образующийся АТФ можно детектировать с помощью фотонов, продуцируемых люциферазой. Таким образом, реакцию секвенирования можно контролировать с помощью системы детектирования люминесценции. Источники возбуждающего излучения, используемые для систем детектирования на основе флуоресценции, не требуются для процедур пиросеквенирования. Подходящие жидкостные системы, детекторы и процедуры, которые можно использовать для применения пиросеквенирования на чипах согласно настоящему раскрытию, описаны, например, в WO 2012/058096, заявке на патент США №2005/0191698 А1, патентах США №7595883 и 7244559.
[0102] В некоторых вариантах реализации можно применять способы, включающие контроль активности ДНК-полимеразы в реальном времени. Например, включение нуклеотидов можно детектировать посредством взаимодействий флуоресцентного резонансного переноса энергии (FRET, от англ. fluorescence resonance energy transfer) между полимеразой, несущей флуорофор, и нуклеотидами, меченными γ-фосфатом, или с помощью волноводов с нулевым режимом. Способы и реагенты для секвенирования на основе FRET раскрыты, например, в Levene et al. Science 299, 682-686 (2003); Lundquist et al. Opt. Lett. 33,1026-1028 (2008); Korlach etal. Proc. Natl. Acad. Sci. USA 105, 1176-1181 (2008).
[0103] Некоторые варианты реализации SBS включают детектирование протона, высвобождающегося при включении нуклеотида в продукт удлинения. Например, для секвенирования на основе детектирования высвобожденных протонов можно применять электрический детектор и связанные с ним способы, которые коммерчески доступны от Ion Torrent (Guilford, СТ, дочерняя компания Life Technologies), или системы и способы секвенирования, раскрытые в патентах США №8262900, 7948015, 8349167 и опубликованной заявке на патент США №2010/0137143 А1.
[0104] Соответственно, в настоящей заявке представлены способы включения аналогов нуклеотидов в ДНК, включающие обеспечение взаимодействия следующих компонентов: (i) измененная полимераза согласно любому из вышеуказанных вариантов реализации, (ii) матрица ДНК; и (iii) раствор нуклеотидов. В некоторых вариантах реализации матрица ДНК включает кластерный чип.В некоторых вариантах реализации нуклеотиды модифицированы по 3'-гидроксилу сахара и включают модификации по 3'-гидроксилу сахара, благодаря чему заместитель имеет больший размер, чем размер встречающейся в природе 3'-гидроксильной группы.
Нуклеиновые кислоты, кодирующие измененные полимеразы
[0105] Настоящее раскрытие также включает молекулы нуклеиновой кислоты, кодирующие измененные полимеразы, описанные в настоящей заявке. Для любой данной измененной полимеразы, которая является мутантной версией полимеразы, для которой известна аминокислотная последовательность и предпочтительно также нуклеотидная последовательность дикого типа, кодирующая полимеразу, можно получить нуклеотидную последовательность, кодирующую мутант, в соответствии с основными принципами молекулярной биологии. Например, учитывая, что нуклеотидная последовательность дикого типа, кодирующая полимеразу 9°N, известна, можно вывести нуклеотидную последовательность, кодирующую любую данную мутантную версию 9°N, имеющую одну или более аминокислотных замен, с применением стандартного генетического кода. Аналогичным образом нуклеотидные последовательности могут быть легко получены для мутантных версий других полимераз, таких как, например, полимераза Vent®, полимераза Deep Vent®, полимераза Pfu, полимераза KOD, полимераза Pab и т.д. Затем можно сконструировать молекулы нуклеиновой кислоты, имеющие требуемую нуклеотидную последовательность, с применением стандартных способов молекулярной биологии, известных в данной области техники.
[0106] В соответствии с вариантами реализации, представленными в настоящей заявке, определенная нуклеиновая кислота включает не только идентичную нуклеиновую кислоту, но также любые незначительные вариации оснований, включая, в частности, замены в случаях, которые приводят к синонимичному кодону (другому кодону, определяющему тот же самый аминокислотный остаток) вследствие вырожденного кода при консервативных аминокислотных заменах. Термин «последовательность нуклеиновой кислоты» также включает последовательность, комплементарную любой одноцепочечной последовательности, приведенной в отношении вариаций оснований.
[0107] Молекулы нуклеиновой кислоты, раскрытые в настоящей заявке, также могут быть преимущественно включены в подходящий вектор экспрессии для экспрессии кодируемых ими белков полимеразы в подходящем хозяине. Включение клонированной ДНК в подходящий вектор экспрессии для последующей трансформации указанной клетки и последующего отбора трансформированных клеток хорошо известно специалистам в данной области техники, как указано в Sambrook et al. (1989), Molecular cloning: A Laboratory Manual, Cold Spring Harbor Laboratory.
[0108] Такой вектор экспрессии включает вектор, содержащий нуклеиновую кислоту в соответствии с представленными в настоящей заявке вариантами реализации, функционально связанную с регуляторными последовательностями, такими как промоторные области, которые способны влиять на экспрессию указанных фрагментов ДНК. Термин «функционально связанный» относится к сопряжению, при котором описанные компоненты находятся во взаимосвязи, позволяющей им функционировать предполагаемым образом. Такие векторы могут быть трансформированы в подходящую клетку-хозяина для обеспечения экспрессии белка в соответствии с представленными в настоящей заявке вариантами реализации.
[0109] Молекула нуклеиновой кислоты может кодировать зрелый белок или белок, имеющий пропоследовательность, в том числе кодирующую лидерную последовательность на белке-предшественнике, который затем расщепляется клеткой-хозяином с образованием зрелого белка. Векторы могут представлять собой, например, плазмидные, вирусные или фаговые векторы, имеющие точку начала репликации и, необязательно, промотор для экспрессии указанного нуклеотида и, необязательно, регулятор промотора. Векторы могут содержать один или более выбираемых маркеров, таких как, например, ген устойчивости к антибиотикам.
[0110] Регуляторные элементы, необходимые для экспрессии, включают промоторные последовательности для связывания РНК-полимеразы и для обеспечения подходящего уровня инициации транскрипции, а также последовательности инициации трансляции для связывания рибосом. Например, бактериальный вектор экспрессии может включать промотор, такой как промотор lac, и для инициации трансляции -последовательность Шайна-Дальгарно и стартовый кодон AUG. Аналогичным образом, эукариотический вектор экспрессии может включать гетерологичный или гомологичный промотор для РНК-полимеразы II, сигнал полиаденилирования, расположенный ниже, стартовый кодон AUG и кодон терминации для отсоединения рибосомы. Такие векторы можно получить коммерчески или собрать из последовательностей, описанных способами, хорошо известными в данной области техники.
[0111] Транскрипция ДНК, кодирующей полимеразу, высшими эукариотами может быть оптимизирована путем включения энхансерной последовательности в вектор. Энхансеры представляют собой цис-действующие элементы ДНК, которые действуют на промотор, повышая уровень транскрипции. Векторы также обычно включают точки начала репликации в дополнение к выбираемым маркерам.
[0112] В настоящем раскрытии также предложен набор для проведения реакции включения нуклеотидов. Набор включает по меньшей мере одну измененную полимеразу, описанную в настоящей заявке, и раствор нуклеотидов в подходящем упаковочном материале в количестве, достаточном для по меньшей мере одной реакции включения нуклеотидов. Необязательно, также включены другие реагенты, такие как буферы и растворы, необходимые для применения измененной полимеразы и раствора нуклеотидов. Также обычно прилагают инструкции по применению упакованных компонентов.
[0113] В некоторых вариантах реализации раствор нуклеотидов включает меченые нуклеотиды. В некоторых вариантах реализации нуклеотиды представляют собой синтетические нуклеотиды. В некоторых вариантах реализации нуклеотиды представляют собой модифицированные нуклеотиды. В некоторых вариантах реализации модифицированный нуклеотид был модифицирован по 3'-гидроксилу сахара, благодаря чему заместитель имеет больший размер, чем встречающаяся в природе 3'-гидроксильная группа. В некоторых вариантах реализации модифицированные нуклеотиды включают модифицированную нуклеотидную или нуклеозидную молекулу, которая включает пуриновое или пиримидиновое основание и рибозный или дезоксирибозный сахарный фрагмент, имеющий ковалентно присоединенную к нему удаляемую З'-ОН блокирующую группу, таким образом, к 3'-атому углерода присоединена группа структуры
 ,
,
где Z представляет собой любой из -C(R')2-O-R'', -C(R')2-N(R'')2, -C(R')2-N(H)R'', -C(R')2-S-R'' и -C(R')2-F,
где каждый R'' представляет собой или является частью удаляемой защитной группы;
каждый R' независимо представляет собой атом водорода, алкил, замещенный алкил, арилалкил, алкенил, алкинил, арил, гетероарил, гетероциклическую, ацильную, циано, алкокси, арилокси, гетероарилокси или амидогруппу или детектируемую метку, присоединенную через связывающую группу; или (R')2 представляет собой алкилиденовую группу формулы =C(R''')2, где каждый R''' может быть одинаковым или различным и выбран из группы, содержащей атомы водорода и галогена и алкильные группы; и
где молекула может реагировать с образованием промежуточного продукта, в котором каждый R'' заменен на Н или, где Z представляет собой -C(R')2-F, F заменен на ОН, SH или NH2, предпочтительно ОН, причем промежуточный продукт диссоциирует в водных условиях с образованием молекулы со свободным 3'ОН;
при условии, что где Z представляет собой -C(R')2-S-R'', обе группы R' не представляют собой Н.
В некоторых вариантах реализации R' модифицированного нуклеотида или нуклеозида представляет собой алкил или замещенный алкил. В некоторых вариантах реализации -Z модифицированного нуклеотида или нуклеозида имеет формулу -C(R')2-N3. В некоторых вариантах реализации Z представляет собой азидометильную группу.
[0114] В некоторых вариантах реализации модифицированные нуклеотиды помечены флуоресцентной меткой, чтобы обеспечить возможность их детектирования. В некоторых вариантах реализации модифицированные нуклеотиды включают нуклеотид или нуклеозид, имеющий основание, присоединенное к детектируемой метке через расщепляемый линкер. В некоторых вариантах реализации детектируемая метка включает в себя флуоресцентную метку.
[0115] Используемое в настоящей заявке выражение «упаковочный материал» относится к одной или более физическим структурам, применяемым для размещения содержимого набора. Упаковочный материал изготавливают известными способами, предпочтительно, чтобы обеспечить стерильную среду, не содержащую загрязняющих веществ. На упаковочном материале присутствует этикетка, на которой указано, что компоненты можно применять для проведения реакции включения нуклеотидов. Кроме того, упаковочный материал содержит инструкции, указывающие, каким образом материалы в наборе применяют для проведения реакции включения нуклеотидов. В настоящей заявке термин «упаковка» относится к твердой матрице или материалу, такому как стекло, пластик, бумага, фольга и тому подобным, способному удерживать полипептиды в установленных границах. «Инструкции по применению» обычно включают конкретное выражение, описывающее концентрацию реагента или по меньшей мере один параметр способа анализа, такой как относительные количества реагента и образца, которые должны быть смешаны, периоды времени выдерживания для смеси реагента/образца, температура, буферные условия и тому подобное.
[0116] Полное раскрытие патентов, патентных документов и публикаций, цитируемых в разделе «Уровень техники изобретения», «Подробное описание изобретения» и где-либо в настоящей заявке, включено в качестве ссылки во всей полноте, как если бы каждый был включен отдельно.
[0117] Обсуждаются иллюстративные варианты реализации настоящего изобретения, и приводится ссылка на возможные вариации в пределах объема настоящего изобретения. Эти и другие вариации, комбинации и модификации в настоящем изобретении будут очевидны специалистам в данной области техники без отклонения от объема изобретения, и следует понимать, что настоящее изобретение не ограничивается иллюстративными вариантами реализации, изложенными в настоящей заявке. Соответственно, настоящее изобретение должно ограничиваться только приведенной ниже формулой изобретения и ее эквивалентами.
Примеры
[0118] Настоящее изобретение проиллюстрировано следующими примерами. Следует понимать, что конкретные примеры, материалы, количества и процедуры следует интерпретировать в широком смысле в соответствии с объемом и сущностью изобретения, изложенными в настоящей заявке.
Пример 1
Общие способы анализа и условия
[0119] Если не указано иное, в настоящей заявке описаны общие условия анализа, используемые в описанных в настоящей заявке примерах.
А. Клонирование и экспрессия полимераз
[0120] Способы получения рекомбинантных нуклеиновых кислот, экспрессии и выделения экспрессированных продуктов известны и описаны в данной области техники. Мутагенез выполняли в кодирующей области, которая кодирует полимеразу 9°N (SEQ ID NO: 1), с применением стандартной методологии сайт-направленного мутагенеза. Подходы на основе ПЦР применяли для амплификации мутировавших кодирующих областей и добавления His-метки. Для каждой выполненной мутации правильную последовательность измененной кодирующей области подтверждали путем определения последовательности клонированной ДНК.
[0121] Кодирующие области мутантной полимеразы, меченные His, были субклонированы в вектор рЕТ11а и трансформированы в экспрессирующие клетки BL21 Star (DE3) (Invitrogen). Ночные культуры из отдельных выбранных колоний применяли для инокуляции экспрессирующих культур в 2,8-литровых колбах. Культуры выращивали при 37°С до OD600 примерно 0,8, затем экспрессию белка индуцировали 0,2 мМ IPTG с последующим 4-часовым дополнительным ростом. Культуры центрифугировали при 7000 об/мин в течение 20 минут.Осадки клеток хранили при -20°С до очистки.
[0122] Осадки размораживали и лизировали 5-кратным масс/об. буфером для лизиса (50 мМ Tris-HCI рН 7,5, 1 мМ ЭДТА, 0,1% ВМЕ и 5% глицерин) в присутствии реагентов Ready-Lyse и Omnicleave (Epicentre) согласно рекомендациям производителя. Конечную концентрацию NaCl повышали до 500 мМ, и лизат инкубировали на льду в течение 5 минут. После центрифугирования супернатант инкубировали при 80°С в течение примерно 70 минут. Всю дальнейшую очистку проводили при 4°С. Супернатант охлаждали в течение 30 минут перед центрифугированием, и очищали с применением колонок 5 мл Ni Sepharose HP (GE). Колонки предварительно уравновешивали буфером А (50 мМ Tris-HCl рН 7,5, 1 мМ ЭДТА, 5% глицерин, 500 мМ NaCl и 20 мМ имидазол). Колонку элюировали градиентом 75 мл от 20 до 500 мМ имидазола. Пиковые фракции объединяли и разбавляли 10% глицерином, чтобы соответствовать проводимости SP буфера А (50 мМ Tris-HCl рН 7,5, 150 мМ NaCl, 1 мМ ЭДТА, 5% глицерин) и загружали в 5 мл колонки SP Sepharose (GE). Колонку элюировали градиентом 100 мл от 150 до 1000 мМ NaCl. Пиковые фракции объединяли, диализовали в буфере для хранения (10 мМ Tris-HCl рН 7,5, 300 мМ KCL, 0,1 мМ ЭДТА и 50% глицерин) и хранили при -20°С.
В. Частота ошибок и анализ фазирования
[0123] Эксперименты по секвенированию применяли для сравнения частоты ошибок и значений фазирования. Если не указано иное, эксперименты проводили на системе MiniSeq™ (Illumina, Inc., Сан-Диего, Калифорния) в соответствии с инструкциями производителя. Например, для каждой полимеразы готовили отдельную смесь для включения (IMX, от англ. incorporation mix) и применяли в коротком прогоне (35 циклов при прочтении 1) или длинном прогоне (227 циклов, из них 151 при прочтении 1 и 76 при прочтении 2). Применяли составы стандартных картриджей с реагентами MiniSeq со средним выходом, при этом стандартную полимеразу заменяли тестируемой полимеразой в концентрации 90 мкг/мл. Время инкубации IMX на проточной кювете варьировали, как указано в приведенных в настоящей заявке примерах. Использованную библиотеку ДНК получали в соответствии со стандартным протоколом TruSeq™ Nano (Illumina, Inc.) с размером целевой вставки 350 п. н. с применением геномной ДНК E. coli; в полученную библиотеку добавляли PhiX ДНК (Illumina, Inc) в молярном соотношении примерно 1:10. Программное обеспечение Illumina RTA применяли для оценки частоты ошибок как для геномов, так и для уровней фазирования.
Пример 2
Эффективность секвенирования измененных полимераз
[0124] Был идентифицирован ряд измененных полимераз, которые имели частоту ошибок и уровни фазирования в коротком прогоне при коротком времени включения, которые не были существенно выше, чем у контрольной полимеразы, применяемой в коротком прогоне при стандартном времени включения. В таблице 2 ниже приведено сравнение эффективности секвенирования измененных полимераз, перечисленных в таблице 1, при времени включения 14 секунд относительно контрольной полимеразы, представленной Pol 812 (SEQ ID NO: 8). Показатели качества, используемые для оценки измененных полимераз, представляли собой частоту фазирования для прочтения 1 («фазирование R1») и частоту накопленных ошибок для контрольных элементов секвенирования E. coli («ошибка Е. coli») и бактериофага PhiX («ошибка PhiX»). Показатели были нормализованы по соответствующему фазированию Pol 812 и частоте ошибок при стандартном (46 секунд) времени включения («812 STD»). Например, частота фазирования R1 Pol 812 при коротком времени включения («812 Fast») в 6 раз выше, чем фазирования R1 при стандартном времени включения, тогда как частота накопленных ошибок E. coli и PhiX выше в 12,5 и 6,4 раз, соответственно. Аналогичным образом, частота фазирования R1 Pol 963 (SEQ ID NO: 9) при коротком времени включения в 5,9 раз выше, чем фазирования R1 Pol 812 при стандартном времени включения, тогда как частота накопленных ошибок E. coli и PhiX для Pol 963 выше в 4,6 и 3,6 раз, соответственно.
[0125] Таблица 2: Показатели эффективности измененных полимераз перечислены в таблице 1.
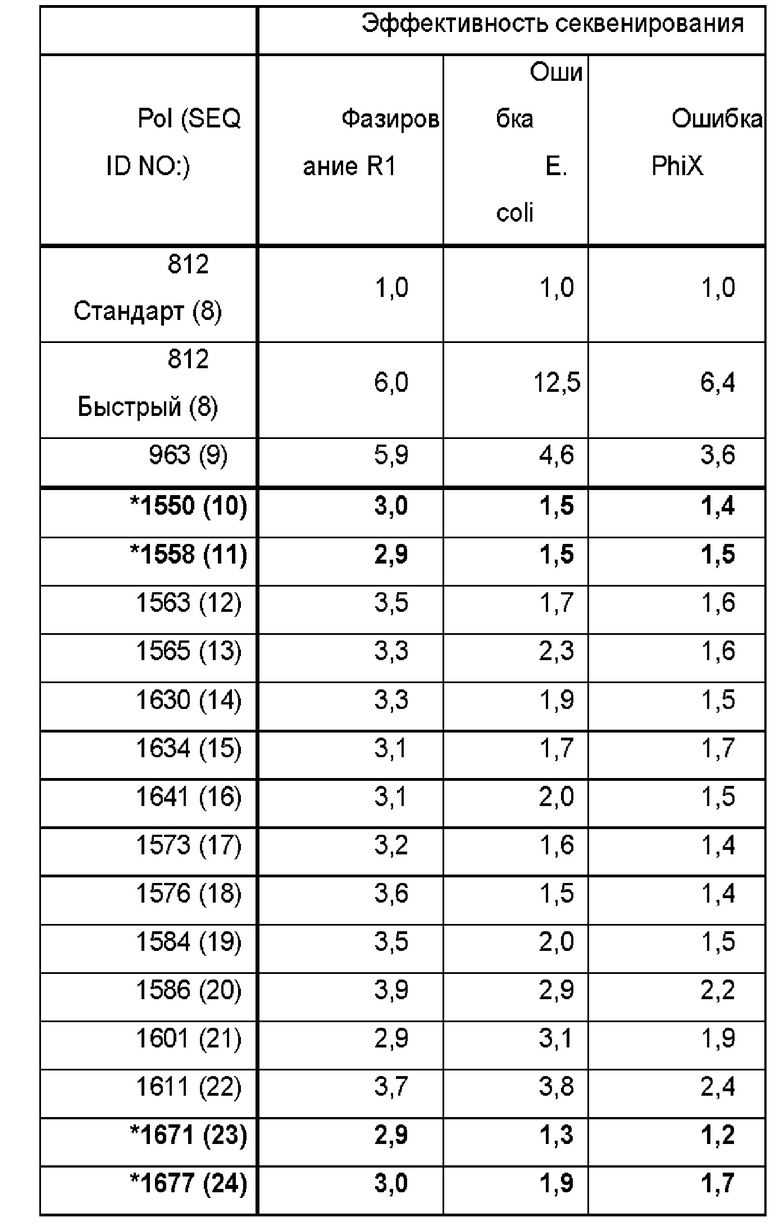
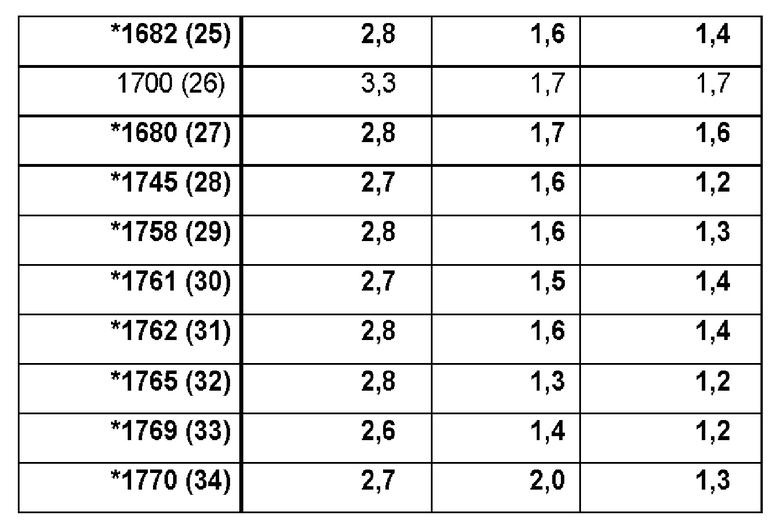
[0126] Измененные полимеразы, характеризующиеся относительными скоростями фазирования не более 3,0 и частотой накопленных ошибок не более 2,0 при коротком времени включения, являются особенно привлекательными кандидатами для применения в быстром SBS. Примеры таких полимераз обозначены жирным шрифтом и звездочкой в Таблице 2. Дополнительные результаты показаны на ФИГ. 2 и 3.
[0127] На ФИГ. 2 показано снижение частоты ошибок фазирования и накопленных ошибок при коротком времени включения, продемонстрированное для одной из измененных полимераз, идентифицированных в примере 2, Pol 1558 (SEQ ID NO: 11), по сравнению с контролем Pol 812 (панели слева). Два фермента демонстрируют сопоставимые частоту фазирования и частоту ошибок при стандартном времени включения (панели справа).
[0128] На ФИГ. 3А показано снижение частоты ошибок фазирования R1 и накопленных ошибок Е. coli при коротком времени включения, продемонстрированное для выбранных измененных полимераз, идентифицированных в примере 2, Pol 1558, Pol 1671 (SEQ ID NO: 23), Pol 1682 (SEQ ID NO: 25) и Pol 1745 (SEQ ID NO: 28), по сравнению с контролями Pol 812 и Pol 963. Пунктирные линии на верхней и нижней панелях показывают накопленную ошибку Е. coli и частоту фазирования R1, продемонстрированную Pol 812 при стандартном времени включения.
[0129] На ФИГ. 3В представлено сравнение частоты фазирования и предварительного фазирования тех же измененных полимераз относительно контролей Pol 812 и Pol 963, демонстрируя снижение частоты фазирования с помощью Pols 1558, 1671, 1682 и 1745.
Пример 3
Активность измененных полимераз в условиях длинного прогона
[0130] Выбранные измененные полимеразы, идентифицированные в примере 2, оценивали с применением различной длины прогонов. Частоту накопленных ошибок PhiX для каждой из измененных полимераз при стандартном времени включения (46 секунд) сравнивали с частотой ошибок фазирования и частотой накопленных ошибок при коротком времени включения (22 секунды) во время длинных прочтений секвенирования (250 циклов при прочтении 1 с последующими 250 циклами при прочтении 2, всего 500 циклов). Условия более длинного прогона приводят к получению большего количества информации о последовательности (т.е. определяют идентичность большего количества нуклеотидов) за прогон, и они являются аналогичными условиям, часто используемым в платформах для секвенирования, например, при секвенировании всего генома. Результаты показаны на ФИГ. 4.
[0131] На ФИГ. 4 представлено сравнение частоты накопленных ошибок PhiX для Pol 1550 (SEQ ID NO: 10) и Pol 1558 (SEQ ID NO: 11) с таковой для контроля Pol 812 (SEQ ID NO: 8) при стандартном и коротком времени включения во время длинных прочтений секвенирования (2×250 циклов). Оба мутанта демонстрируют заметное снижение частоты ошибок после выполнения секвенирования спаренных концов. Кроме того, Pol 1550 показывает значительное снижение частоты ошибок по сравнению с Pol 812 во время первого прочтения секвенирования.
Пример 4
Оценка параметров секвенирования на NovaSeq™
[0132] Одну из измененных полимераз, идентифицированных в примере 2, Pol 1671, оценивали на платформе Illumina NovaSeq™ с применением проточной кюветы S1 и химии секвенирования NovaSeq™. Частота ошибок, уровни фазирования и другие параметры качества секвенирования были определены для прочтений 1 и 2 при коротком и стандартном времени включения. Результаты для Pol 1671 и Pol 812 обобщены на ФИГ. 5.
[0133] На ФИГ. 5А показано сравнение показателей секвенирования NovaSeq™ для Pol 1671 (SEQ ID NO: 23), продемонстрированных при коротких временах включения (10 сек), с таковыми для контроля Pol 812 (SEQ ID NO: 8), продемонстрированных при стандартном (40 сек) и коротком (10 сек) времени включения. Верхние панели демонстрируют процент кластеров, прошедших фильтр («Clusters PF»); нижние панели демонстрируют частоту накопленных ошибок PhiX. Светлые незаштрихованные кружки обозначают показатели Pol 812 при стандартном времени включения, тогда как темные незаштрихованные кружки обозначают показатели Pol 812 при коротком времени включения. Все показатели Pol 1671, обозначенные заштрихованными кружками, имеют короткое время включения. Результаты показывают, что Pol 1671 демонстрирует сопоставимую эффективность в обоих прочтениях при коротком времени включения с таковой для Pol 812 при стандартном времени включения.
[0134] На ФИГ. 5В обобщены частоты накопленных ошибок PhiX, значения Q30 и частоты фазирования, которые демонстрирует Pol 1671 по сравнению с контролем Pol 812 для прочтений 1 и 2 NovaSeq™ при стандартном (40 сек) и коротком (22 сек) времени включения. Значительное улучшение качества секвенирования для обоих прочтений наблюдалось при применении Pol 1671.
[0135] Полное раскрытие всех патентов, патентных заявок и публикаций, а также материалов, доступных в электронном виде (включая, например, предоставление нуклеотидных последовательностей, например, в GenBank и RefSeq, и предоставление аминокислотных последовательностей, например, в SwissProt, PIR, PRF, PDB, и трансляции из аннотированных кодирующих областей в GenBank и RefSeq), цитируемые в настоящей заявке, полностью включены в настоящую заявку в качестве ссылки. Дополнительные материалы, на которые присутствуют ссылки в публикациях (такие как дополнительные таблицы, дополнительные фигуры, дополнительные материалы и способы и/или дополнительные экспериментальные данные), также включены в настоящую заявку в качестве ссылки во всей их полноте. В случае, если существует какое-либо несоответствие между раскрытием настоящей заявки и раскрытием (раскрытиями) любого документа, включенного в настоящую заявку посредством ссылки, раскрытие настоящей заявки имеет преимущественную силу. Вышеприведенное подробное раскрытие и примеры приведены только для ясности понимания. Из этого не следует понимать никаких излишних ограничений. Настоящее изобретение не ограничивается точными деталями, показанными и описанными, поскольку варианты, очевидные для специалиста в данной области техники, будут включены в настоящее изобретение, определенное формулой изобретения.
[0136] Если не указано иное, все числа, выражающие количества компонентов, молекулярные массы и так далее, используемые в описании и формуле изобретения, следует понимать как измененные во всех случаях термином «примерно». Соответственно, если не указано иное, числовые параметры, изложенные в описании и формуле изобретения, являются приближениями, которые могут варьироваться в зависимости от желаемых свойств, которые стремятся получить с помощью настоящего изобретения. По меньшей мере, и не в качестве попытки ограничить доктрину эквивалентов объемом формулы изобретения, каждый числовой параметр должен, по меньшей мере, быть истолкован в свете количества приведенных значащих цифр и с применением обычных методов округления.
[0137] Несмотря на то, что числовые диапазоны и параметры, определяющие широкий объем изобретения, являются приблизительными, числовые значения, изложенные в конкретных примерах, указаны с максимально возможной точностью. Однако все числовые значения по своей сути содержат диапазон, обязательно являющийся результатом стандартного отклонения, обнаруженного в соответствующих испытательных измерениях.
[0138] Все заголовки предназначены для удобства читателя и не должны использоваться для ограничения смысла текста, следующего за заголовком, если не указано иное.





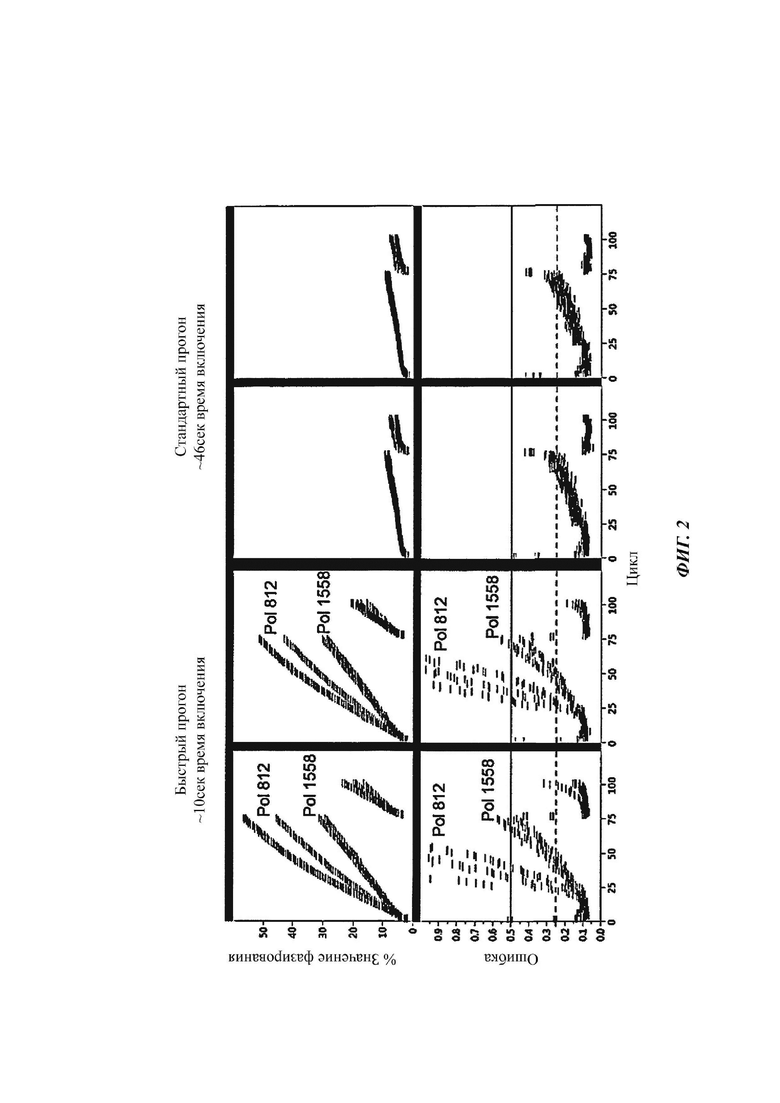
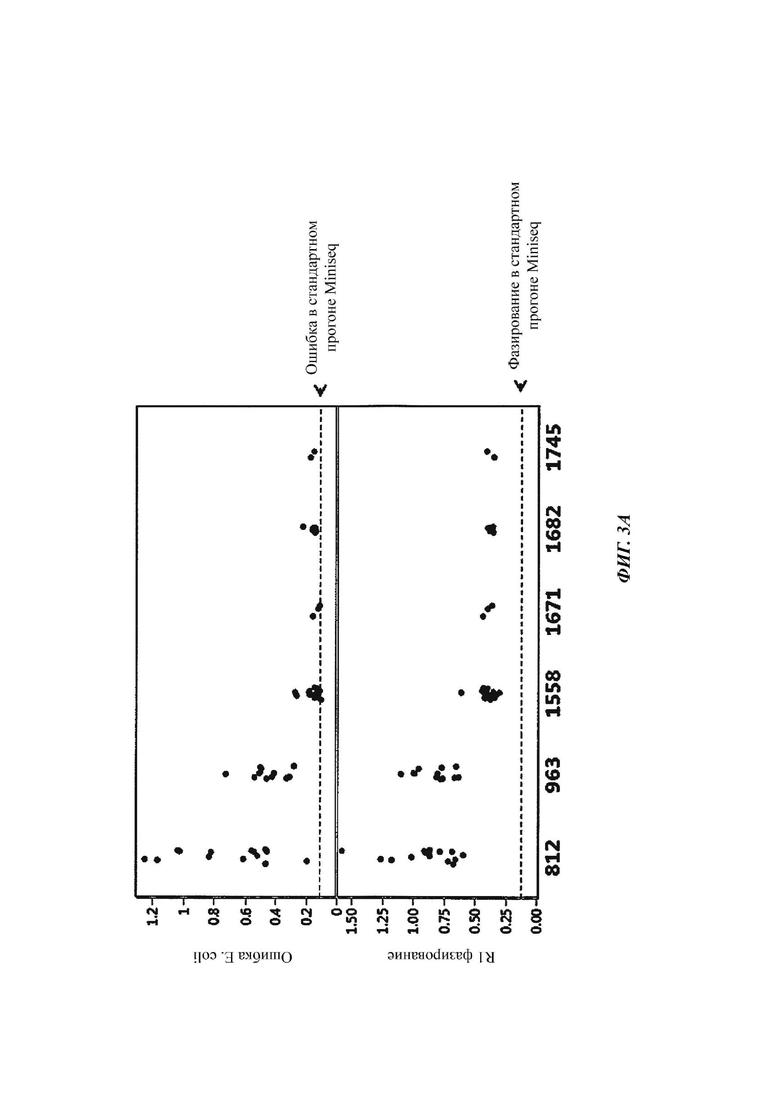

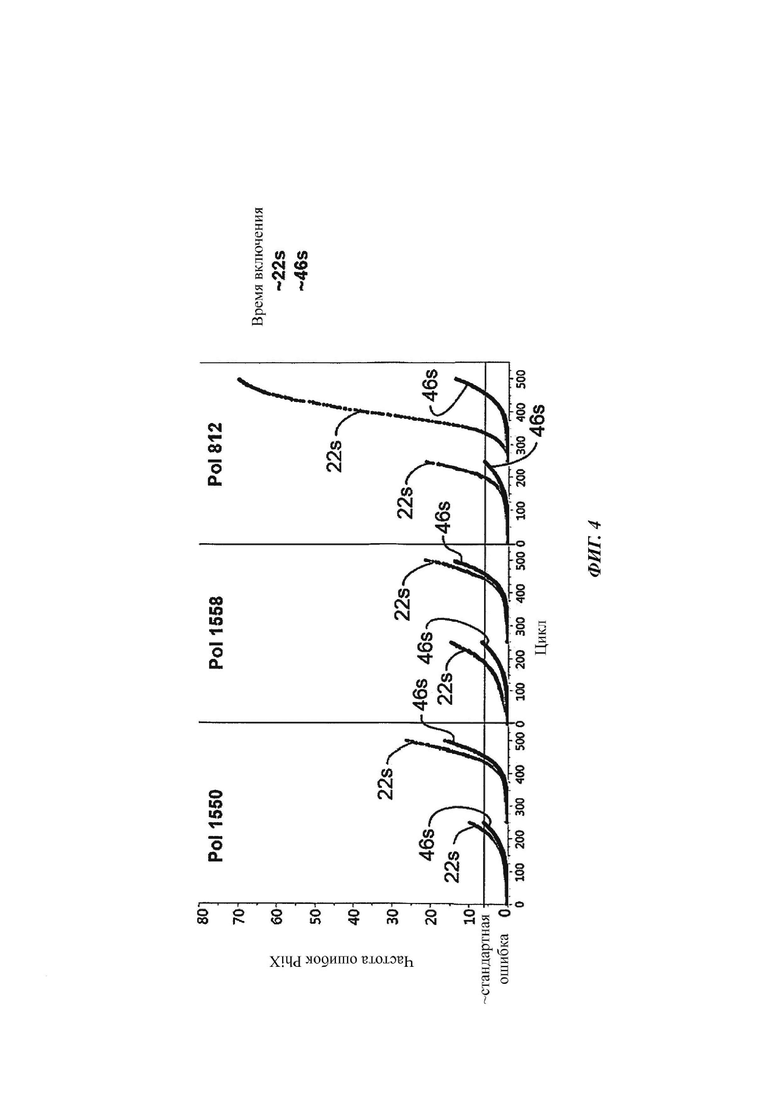
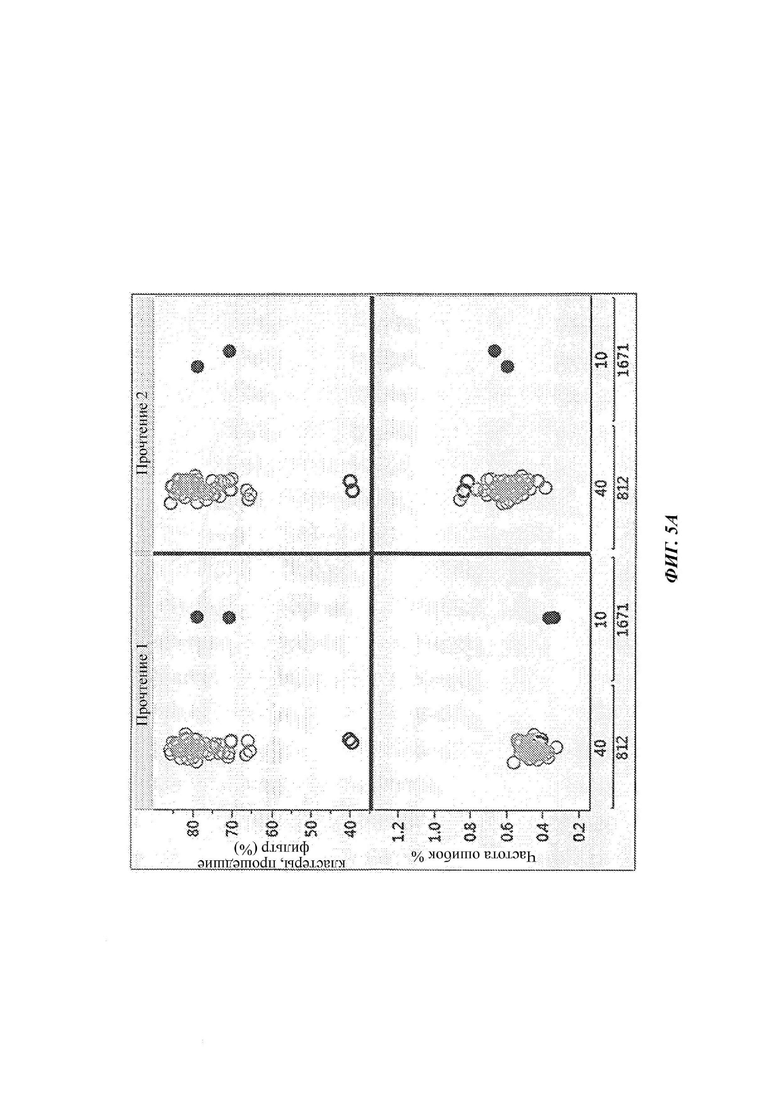
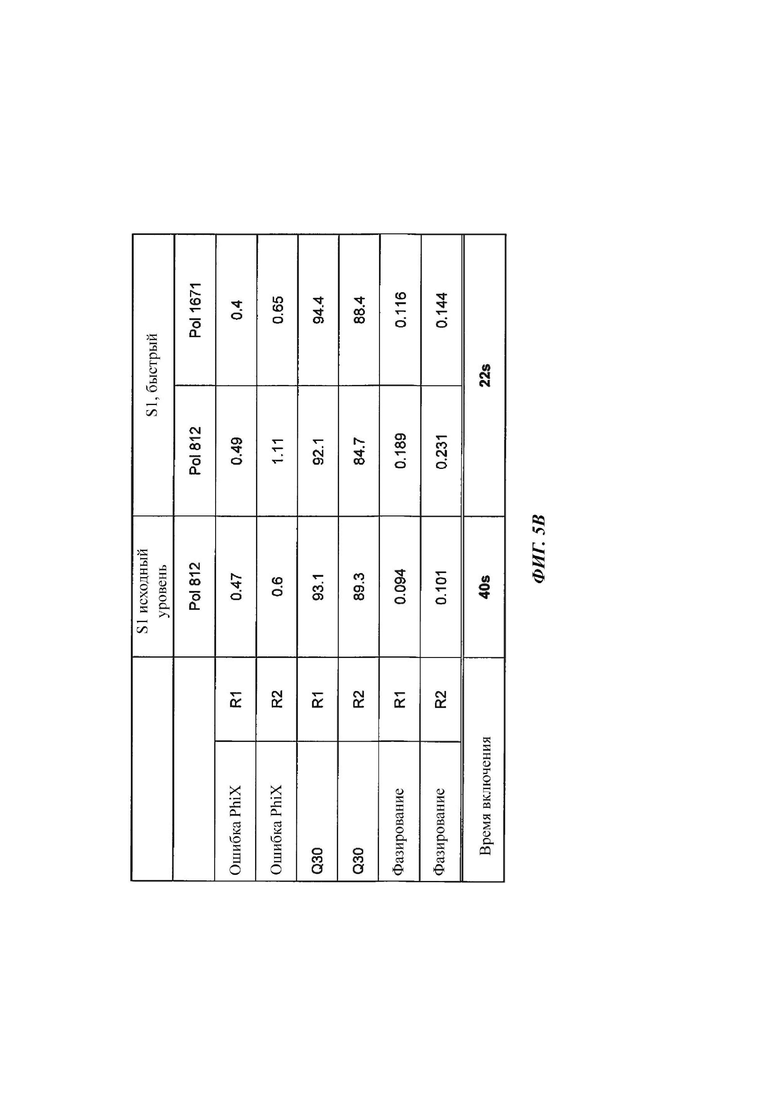
Изобретение относится к биотехнологии и представляет собой модифицированные ферменты полимеразы для улучшенного включения нуклеотидов и аналогов нуклеотидов, в частности измененные полимеразы, которые сохраняют высокую достоверность при сокращении времени включения, а также способы и наборы, в которых их применяют. 9 н. и 47 з.п. ф-лы, 5 ил., 2 табл., 4 пр.
1. Рекомбинантная ДНК-полимераза, содержащая аминокислотную последовательность, которая по меньшей мере на 80% идентична аминокислотной последовательности SEQ ID NO: 1 ДНК-полимеразы 9°N, при этом рекомбинантная ДНК-полимераза содержит мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, и по меньшей мере две мутации по типу замены аминокислоты в положениях, функционально эквивалентных Phe152, Met329 или Val471 в аминокислотной последовательности ДНК-полимеразы 9°N.
2. Полимераза по п. 1, где мутация по типу замены в положении, функционально эквивалентном Tyr497, содержит мутацию до неполярной, гидрофобной или незаряженной аминокислоты.
3. Полимераза по п. 1, где мутация по типу замены в положении, функционально эквивалентном Tyr497, содержит мутацию до Gly.
4. Полимераза по п. 1, где мутация по типу замены в положении, функционально эквивалентном Phe152, содержит мутацию до неполярной, гидрофобной или незаряженной аминокислоты.
5. Полимераза по п. 1, где мутация по типу замены в положении, функционально эквивалентном Phe152, содержит мутацию до Gly.
6. Полимераза по п. 1, где полимераза дополнительно содержит мутацию по типу замены в положении, функционально эквивалентном Val278, где указанная мутация по типу замены содержит мутацию до неполярной или гидрофобной аминокислоты.
7. Полимераза по п. 1, где полимераза дополнительно содержит мутацию по типу замены в положении, функционально эквивалентном Val278, где указанная мутация по типу замены содержит мутацию до Leu.
8. Полимераза по п. 1, где мутация по типу замены в положении, функционально эквивалентном Met329, содержит мутацию до полярной аминокислоты.
9. Полимераза по п. 1, где мутация по типу замены в положении, функционально эквивалентном Met329, содержит мутацию до His.
10. Полимераза по п. 1, где мутация по типу замены в положении, функционально эквивалентном Val471, содержит мутацию до полярной или незаряженной аминокислоты.
11. Полимераза по п. 1, где мутация по типу замены в положении, функционально эквивалентном Val471, содержит мутацию до Ser.
12. Полимераза по п. 1, где полимераза дополнительно содержит мутацию по типу замены в положении, функционально эквивалентном Thr514, где указанная мутация по типу замены содержит мутацию до неполярной или гидрофобной аминокислоты.
13. Полимераза по п. 1, где полимераза дополнительно содержит мутацию по типу замены в положении, функционально эквивалентном Thr514, где указанная мутация по типу замены содержит мутацию до Ala.
14. Полимераза по п. 1, где полимераза дополнительно содержит мутацию по типу замены в положении, функционально эквивалентном Leu631, где указанная мутация по типу замены содержит мутацию до неполярной или гидрофобной аминокислоты.
15. Полимераза по п. 1, где полимераза дополнительно содержит мутацию по типу замены в положении, функционально эквивалентном Leu631, где указанная мутация по типу замены содержит мутацию до Met.
16. Полимераза по п. 1, где полимераза дополнительно содержит мутацию по типу замены в положении, функционально эквивалентном Glu734, где указанная мутация по типу замены содержит мутацию до полярной аминокислоты.
17. Полимераза по п. 1, где полимераза дополнительно содержит мутацию по типу замены в положении, функционально эквивалентном Glu734, где указанная мутация по типу замены содержит мутацию до Arg.
18. Полимераза по п. 1, где указанная полимераза содержит по меньшей мере две, по меньшей мере три, по меньшей мере четыре, по меньшей мере пять, по меньшей мере шесть или семь мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Phe152, Val278, Met329, Val471, Thr514, Leu631 и Glu734 в аминокислотной последовательности ДНК-полимеразы 9°N.
19. Полимераза по любому из пп. 1-18, где полимераза дополнительно содержит мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
20. Рекомбинантная ДНК-полимераза, содержащая аминокислотную последовательность, которая по меньшей мере на 80% идентична аминокислотной последовательности SEQ ID NO: 1 ДНК-полимеразы 9°N, при этом рекомбинантная ДНК-полимераза содержит мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, и по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Arg247, Glu599, Lys620, His633 или Val661 в аминокислотной последовательности ДНК-полимеразы 9°N.
21. Полимераза по п. 20, где мутация по типу замены в положении, функционально эквивалентном Arg247, содержит мутацию до неполярной или незаряженной аминокислоты.
22. Полимераза по п. 20, где мутация по типу замены в положении, функционально эквивалентном Arg247, содержит мутацию до Tyr.
23. Полимераза по п. 20, где мутация по типу замены в положении, функционально эквивалентном Glu599, содержит мутацию до полярной аминокислоты.
24. Полимераза по п. 20, где мутация по типу замены в положении, функционально эквивалентном Glu599, содержит мутацию до Asp.
25. Полимераза по п. 20, где мутация по типу замены в положении, функционально эквивалентном Lys620, содержит мутацию до полярной аминокислоты.
26. Полимераза по п. 20, где мутация по типу замены в положении, функционально эквивалентном Lys620, содержит мутацию до Arg.
27. Полимераза по п. 20, где мутация по типу замены в положении, функционально эквивалентном His633, содержит мутацию до неполярной, гидрофобной или незаряженной аминокислоты.
28. Полимераза по п. 20, где мутация по типу замены в положении, функционально эквивалентном His633, содержит мутацию до Gly.
29. Полимераза по п. 20, где мутация по типу замены в положении, функционально эквивалентном Val661, содержит мутацию до полярной аминокислоты.
30. Полимераза по п. 20, где мутация по типу замены в положении, функционально эквивалентном Val661, содержит мутацию до Asp.
31. Полимераза по п. 20, где полимераза содержит по меньшей мере две, по меньшей мере три, по меньшей мере четыре или пять мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Arg247, Glu599, Lys620, His633 и Val661 в аминокислотной последовательности ДНК-полимеразы 9°N.
32. Полимераза по любому из пп. 20-31, где полимераза дополнительно содержит мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
33. Рекомбинантная ДНК-полимераза, содержащая аминокислотную последовательность, которая по меньшей мере на 80% идентична аминокислотной последовательности SEQ ID NO: 1 ДНК-полимеразы 9°N, при этом рекомбинантная ДНК-полимераза содержит (i) мутацию по типу замены аминокислоты в положении, функционально эквивалентном Tyr497, (ii) по меньшей мере две мутации по типу замены аминокислоты в положениях, функционально эквивалентных Phe152, Met329 или Val471 в аминокислотной последовательности ДНК-полимеразы 9°N, и (iii) по меньшей мере одну мутацию по типу замены аминокислоты в положении, функционально эквивалентном Arg247, Glu599, Lys620, His633 или Val661 в аминокислотной последовательности ДНК-полимеразы 9°N.
34. Полимераза по п. 33, где указанная полимераза содержит по меньшей мере три, по меньшей мере четыре, по меньшей мере пять, по меньшей мере шесть или семь мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Phe152, Val278, Met329, Val471, Thr514, Leu631 и Glu734 в аминокислотной последовательности ДНК-полимеразы 9°N.
35. Полимераза по п. 33, где полимераза содержит по меньшей мере две, по меньшей мере три, по меньшей мере четыре или пять мутаций по типу замены аминокислоты в положениях, функционально эквивалентных аминокислоте, выбранной из Arg247, Glu599, Lys620, His633 или Val661 в аминокислотной последовательности ДНК-полимеразы 9°N.
36. Полимераза по любому из пп. 33-35, где полимераза дополнительно содержит мутации по типу замены аминокислоты в положениях, функционально эквивалентных аминокислотам Met129, Asp141, Glu143, Cys223, Leu408, Tyr409, Pro410 и Ala485 в аминокислотной последовательности ДНК-полимеразы 9°N.
37. Полимераза по любому из пп. 1-36, где указанная полимераза представляет собой ДНК-полимеразу семейства типа В.
38. Рекомбинантная ДНК-полимераза, содержащая аминокислотную последовательность согласно любой из SEQ ID NO: 10-34.
39. Молекула нуклеиновой кислоты, кодирующая полимеразу, как определено в любом из пп. 1-36 и 38.
40. Вектор экспрессии, содержащий молекулу нуклеиновой кислоты по п. 39.
41. Клетка-хозяин, содержащая вектор по п. 40, где указанная клетка-хозяин экспрессирует белок, кодируемый вектором экспрессии по п. 40.
42. Способ включения модифицированных нуклеотидов в растущую цепь ДНК, включающий обеспечение взаимодействия следующих компонентов: (i) полимераза по любому из пп. 1-36 и 38, (ii) матрица ДНК и (iii) раствор нуклеотидов.
43. Способ по п. 42, где матрица ДНК содержит кластерный чип.
44. Набор для проведения реакции включения нуклеотидов, содержащий: полимеразу, как определено в любом из пп. 1-36 и 38, и раствор нуклеотидов.
45. Набор по п. 44, где раствор нуклеотидов содержит меченые нуклеотиды.
46. Набор по п. 44, где нуклеотиды содержат синтетические нуклеотиды.
47. Набор по п. 44, где нуклеотиды содержат модифицированные нуклеотиды.
48. Набор по п. 44, где модифицированные нуклеотиды модифицированы по 3'-гидроксилу сахара, благодаря чему заместитель имеет больший размер, чем встречающаяся в природе 3'-гидроксильная группа.
49. Набор по п. 47, где модифицированные нуклеотиды содержат модифицированную нуклеотидную или нуклеозидную молекулу, которая содержит пуриновое или пиримидиновое основание и рибозный или дезоксирибозный сахарный фрагмент, имеющий ковалентно присоединенную к нему удаляемую 3'-ОН блокирующую группу, таким образом, к 3'-атому углерода присоединена группа структуры
 ,
,
при этом Z представляет собой любой из -C(R')2-O-R'', -C(R')2-N(R'')2, -C(R')2-N(H)R'', -C(R')2-S-R'' и -C(R')2-F, при этом каждый R'' представляет собой или является частью удаляемой защитной группы;
каждый R' независимо представляет собой атом водорода, алкил, замещенный алкил, арилалкил, алкенил, алкинил, арил, гетероарил, гетероциклическую, ацильную, циано, алкокси, арилокси, гетероарилокси или амидогруппу или детектируемую метку, присоединенную через связывающую группу; или (R')2 представляет собой алкилиденовую группу формулы =C(R''')2, при этом каждый R''' может быть одинаковым или различным и выбран из группы, включающей атомы водорода и галогена и алкильные группы; и
при этом указанная молекула может реагировать с образованием промежуточного продукта, в котором каждый R'' заменен на Н, или при этом Z представляет собой -C(R')2-F, F заменен на ОН, SH или NH2, предпочтительно ОН, причем промежуточный продукт диссоциирует в водных условиях с образованием молекулы со свободным 3'ОН;
при условии, что где Z представляет собой -C(R')2-S-R'', обе группы R' не представляют собой Н.
50. Набор по п. 49, где R' модифицированного нуклеотида или нуклеозида представляет собой алкил или замещенный алкил.
51. Набор по п. 49, где -Z модифицированного нуклеотида или нуклеозида имеет формулу -C(R')2-N3.
52. Набор по п. 51, где Z представляет собой азидометильную группу.
53. Набор по п. 47, где модифицированные нуклеотиды помечены флуоресцентной меткой, чтобы обеспечить возможность их детектирования.
54. Набор по п. 47, где модифицированные нуклеотиды содержат нуклеотид или нуклеозид, имеющий основание, присоединенное к детектируемой метке через расщепляемый линкер.
55. Набор по п. 54, где детектируемая метка содержит флуоресцентную метку.
56. Набор по п. 44, дополнительно содержащий одну или более молекул ДНК матрицы и/или праймеры.
| WO 2006037064 A3, 09.04.2009 | |||
| Устройство для закрепления лыж на раме мотоциклов и велосипедов взамен переднего колеса | 1924 |
|
SU2015A1 |
| Способ получения цианистых соединений | 1924 |
|
SU2018A1 |
| Воздушно-механическая форсунка | 1928 |
|
SU11362A1 |
Авторы
Даты
2022-09-12—Публикация
2019-10-31—Подача