Настоящая заявка испрашивает приоритет согласно предварительной заявке на патент США номер 61/867224, поданной 19 августа 2013 г., которая полностью включена в настоящую заявку посредством ссылки.
ОБЛАСТЬ ТЕХНИКИ
Согласно настоящему изобретению предложена технология, которая относится к секвенированию нового поколения и, в частности, к способам, композициям, наборам и системам для получения библиотеки для секвенирования нового поколения, содержащей перекрывающиеся фрагменты ДНК, и применению указанной библиотеки для секвенирования одной или более нуклеиновых кислот-мишеней, но не ограничивается указанными.
УРОВЕНЬ ТЕХНИКИ
Последовательности нуклеиновых кислот кодируют информацию, необходимую для функционирования и воспроизведения живых организмов. Таким образом, определение таких последовательностей является полезным инструментом для фундаментальных исследований, связанных с изучением образа жизни и места обитания организмов, а также для прикладных исследований, таких как разработка лекарственных средств. Инструменты секвенирования используются в медицине для диагностики и разработки схем лечения многих патологических состояний, включая рак, инфекционные заболевания, заболевания сердца, аутоиммунные расстройства, множественный склероз и ожирение. В промышленности секвенирование используется для разработки усовершенствованных ферментативных процессов и синтетических организмов. В биологии такие инструменты используются для исследования, например, состояния экосистем, и таким образом, имеют широкий диапазон применения.
Одним ключевым моментом индустрии секвенирования стал поиск технологий секвенирования нуклеиновых кислот с более высокой производительностью и/или более низкой стоимостью, которые иногда называют технологиями секвенирования «нового поколения» («next generation» sequencing, NGS). При разработке технологий секвенирования с более высокой производительностью и/или с более низкой стоимостью цель заключается в создании более удобной технологии для секвенирования. Указанных целей можно достигнуть с помощью использования платформ и способов секвенирования, обеспечивающих получение образца с достижением большего количества образцов значительной сложности, секвенирования большего количества сложных образцов и/или обеспечения большого объема получения и анализа информации за короткий период времени. Различные способы, такие как, например, секвенирование путем синтеза, секвенирование путем гибридизации и секвенирование путем лигирования, совершенствуют для решения указанных задач.
Доступно большое количество платформ для секвенирования нового поколения (NGS) для высокоэффективного массового параллельного секвенирования нуклеиновых кислот. Во многих из указанных систем, таких как системы HiSeq и MiSeq, полученных компанией Illumina, используется способ секвенирования путем синтеза (sequencing-by-synthesis, SBS), согласно которому нуклеотидную последовательность определяют путем выявления и идентификации последовательно каждого основания. При использовании указанного конкретного способа для идентификации 1 основания требуется 1 цикл химического процесса SBS (который может включать четыре отдельные реакции, разделенные этапами отмывки).
В настоящее время указанные технологии обеспечивают максимальную достижимую длину прочтения, составляющую ~250 оснований, которую можно довести до ~400 оснований (2×250 оснований с достаточным перекрыванием для сборки), если два прочтения со спаренными концами высокого качества получают и собирают на основе одной и той же матрицы. Осуществление каждого SBS цикла занимает приблизительно 4 минуты; таким образом, при использовании способа спаренных концов для получения информации последовательности размером ~400 оснований осуществление 500 циклов SBS, необходимых для получения двух прочтений, составляющих ~250 оснований, занимает приблизительно 37 часов. Кроме того, производительность и качество большинства технологий циклического секвенирования по существу снижается после определения ~100 оснований, что приводит к появлению степени неопределенности, связанной с отдельными прочтениями последовательности, составляющими более ~100 оснований в длину, и более длинными последовательностями, в которых они используются. Из-за указанных качественных и временных ограничений современных платформ NGS нарастающая потребность в длинных нуклеотидных последовательностях высокого качества приводит к предельной нагрузке установленного базового оборудования устройства для секвенирования. В результате существует необходимость в технологиях, которые обеспечивают последовательности высокого качества, состоящие из ~500 оснований или более при более коротком времени осуществления секвенирования, занимающим несколько часов вместо нескольких дней.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
При попытках получить более длинные последовательности с помощью технологии NGS применяли подход сборки множества коротких прочтений для получения более длинной последовательности. Например, согласно технологии Moleculo, предложенной компанией Illumina, сначала выделяют одну копия длинного (~10 т.п.н.) фрагмента ДНК. Указанный длинный фрагмент ДНК клонально амплифицируют и затем фрагментируют с получением более мелких фрагментов, составляющих приблизительно 300-800 оснований. В конечном итоге адаптеры со штрихкодами присоединяют к указанным более мелким фрагментам с использованием трнаспозазы для создания библиотеки для секвенирования. Стандартный протокол SBS используют для получения ~300-500 оснований последовательности на основе матрицы-мишени (2×150 оснований или 2×250 оснований) и после получения последовательностей штрихкоды используют для анализа и сборки прочтений для получения последовательности оригинальной ДНК, составляющей ~10 т.п.н. Другой способ включает создание библиотеки перекрывающихся фрагментов, подходящей для секвенатора Illumina, который обеспечивает прочтения размером от ~400 до 460 оснований путем сборки двух прочтений размером ~250 оснований, которые перекрываются на ~20-50 оснований (см., например, Lundin, et al. (2012) Scientific Reports 3: 1186). Указанная библиотека перекрывающихся фрагментов создается в основном путем мечения фрагментов с помощью специфичных адаптерных последовательностей с последующими этапом расщепления и выбора точно определенного размера.
Соответственно, согласно настоящей заявке, предложена технология для секвенирования, в которой используются относительно короткие прочтения (например, содержащие менее чем 300 или менее чем 200 оснований, например, ~30-50 оснований) для достижения длинной непрерывной последовательности высокого качества, сравнимой или улучшенной по сравнению со стандартными технологиями. В отличие от стандартных технологий для осуществления предложенной технологии требуется только короткий период времени (например, ~3-4 часов) работы на секвенаторе (например, платформе MiSeq, Illumina), что, таким образом, существенно снижает время использования устройства для секвенирования, необходимого для выполнения цикла секвенирования. Более того, технология приводит к получению более длинных последовательностей (например, от ~500 п.о. до 1000 п.о.) или более последовательностей более высокого качества по сравнению со стандартной технологией. Также время выполнения не увеличивается в зависимости от размера нуклеиновой кислоты, подлежащей секвенированию, так как размер коротких прочтений (например, ~30-50) остается таким же независимо от размера нуклеиновой кислоты, подлежащей секвенированию.
Настоящая технология не ограничивается какими-либо конкретными платформами для секвенирования и является общеприменимой и не зависит от платформы. Например, помимо снижения времени выполнения на системах Illumina, сходное уменьшение времени наблюдается при получении последовательностей с использованием, например, систем Ion Torrent, Life Technologies, и GeneReader, Qiagen. В частности, тогда как получение последовательности, состоящей из ~400 оснований, с использованием стандартного способа получения образца с помощью системы Ion Torrent и технологии секвенирования занимает приблизительно 4 часа, технология, предложенная в настоящей заявке, обеспечивает уменьшение этого времени до приблизительно 20-30 минут. Согласно некоторым вариантам реализации изобретения, технология применима для способов на основе эмульсионной ПЦР, способов на основе гранул и способов не на основе гранул, и, таким образом, может применяться в системах SOLiD, Life Technologies и платформах секвенирования NGS, Qiagen.
Указанная технология обеспечивает последовательность высокого качества при сниженном времени секвенирования по сравнению со стандартными технологиями. Технология не зависит от платформы и, таким образом, совместима с существующими устройствами для секвенирования. Технология, согласно некоторым вариантам реализации изобретения, улучшает существующие платформы для NGS путем, например, повышения длины прочтения для существующей платформы и уменьшения времени получения последовательности. Более того, дополнительное преимущество настоящей технологии заключается в том, что она снижает использованием дорогих реагентов для секвенирования и, таким образом, может снижать общую стоимость секвенирования из расчета на одно основание.
Кратко, технология включает получение набора определенных перекрывающихся коротких фрагментов библиотеки последовательностей (например, содержащих менее чем 300 или менее чем 200 оснований, например, ~30-50 оснований), покрывающих область нуклеиновой кислоты, подлежащую секвенированию, и смещенных друг относительно друга, например, на 1-20, 1-10 или 1-5 оснований (например, согласно некоторым вариантам реализации изобретения, на 1 основание). После создания набора последовательностей с использованием перекрывающихся библиотек используют биоинформационный алгоритм сборки для «сшивки» набора следующих друг за другом коротких перекрывающихся последовательностей с получением последовательности нуклеиновой кислоты.
Во-первых, качество последовательности является высоким, поскольку каждое основание в нуклеиновой кислоте, подлежащей секвенированию, секвенируют с высоким покрытием (например, 10-кратным - 1000-кратным покрытием, например, от 50-кратного до 500-кратного покрытия) в зависимости от длины полученных коротких последовательностей и смещения соседних покрывающих последовательностей по отношению друг к другу. Высокая частота отбора проб для каждого основания минимизирует или устраняет ошибки секвенирования с обеспечения большего количества информации для процесса сборки, который определяет консенсусную идентичность каждого основания. Кроме того, первое основание (например, первые ~20-100 оснований), определенное в ходе рабочего цикла секвенирования, в целом имеет лучшее качество. Таким образом, при использовании указанных первых оснований, определенных во время первого этапа каждого рабочего цикла секвенирования (например, первых ~30-50 оснований), информации последовательности высокого качества используется в сборке последовательности. Технология, таким образом, приводит к минимизации ошибок секвенирования, в частности, в способах применения, при которых являются желательными длинные прочтения последовательности, сохраняющие информацию фазирования и сцепления, связанную с прочтениями и последовательностями.
Во-вторых, время работы секвенатора снижается из-за того, что для определения каждой короткой последовательности (например, размером ~30-50 оснований) требуется небольшое количество циклов секвенирования (например, 1 цикл на основание, например, ~30-50 циклов) на устройстве для секвенирования. Благодаря параллельному определению всех коротких последовательностей в наборе время секвенирования, необходимое для получения последовательности нуклеиновой кислоты, подлежащей секвенированию, значительно снижается, например, от одной восьмой до одной десятой времени, необходимого для получения той же нуклеиновой кислоты, подлежащей секвенированию, с помощью стандартных технологий для секвенирования.
Указанная технология для получения библиотеки NGS, секвенирования и последующего анализа и сборки коротких прочтений обеспечивает получение непрерывной последовательности высокого качества с сохраненной информацией фазирования, составляющей более чем ~500 п.о. (например, 600, 700, 800 п.о. или более). Технология применяется, например, для секвенирования неизвестных участков, начиная с известной области, например, для поиска структурных вариантов, таких как генные транслокации, например, делеции, и идентификации неизвестных партнеров слияния генов. Более того, технология улучшает способности существующих платформ для секвенирования NGS, связанные с длиной прочтения, временем рабочего цикла и стоимостью, без каких-либо обновлений и/или изменений в существующем установленном оборудовании и существующих химических реакциях секвенирования.
Согласно некоторым вариантам реализации изобретения, технология относится к способу определения нуклеотидной последовательности-мишени, включающему определение первой нуклеотидной субпоследовательности нуклеотидной последовательности-мишени, где 5' конец указанной первой нуклеотидной субпоследовательности находится на нуклеотиде x1 нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде y1 нуклеотидной последовательности-мишени; определение второй нуклеотидной субпоследовательности нуклеотидной последовательности-мишени, где 5' конец указанной второй нуклеотидной субпоследовательности находится на нуклеотиде х2 нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде у2 нуклеотидной последовательности-мишени; сборку первой нуклеотидной субпоследовательности и второй нуклеотидной субпоследовательности для получения консенсусной последовательности для нуклеотидной последовательности-мишени, где х2<у1; и (y1-x1)<100, (у2-х2)<100, и (у2-y1)<5. Согласно некоторым вариантам реализации изобретения, длина фрагментов составляет менее чем 100 п.о., менее чем 90 п.о., менее чем 80 п.о., менее чем 70 п.о., менее чем 60 п.о., менее чем 55 п.о., менее чем 50 п.о., менее чем 45 п.о., менее чем 40 п.о. или менее чем 35 п.о. Соответственно, согласно некоторым вариантам реализации изобретения, (y1-x1)<100, 90, 80, 70, 60, 55, 50, 45, 40 или 35 и (у2-х2)<100, 90, 80, 70, 60, 55, 50, 45, 40 или 35. Согласно некоторым вариантам реализации изобретения, длина фрагменты составляет менее чем 50 п.о.; соответственно, согласно некоторым вариантам реализации изобретения, (y1-x1)<50, и (у2-х2)<50.
Согласно некоторым вариантам реализации изобретения, 3' концы фрагментов отличаются по сравнению с последовательностью-мишенью менее чем на 4 или менее чем на 3 основания; соответственно, согласно некоторым вариантам реализации изобретения, (у2-y1)<4, или (у2-y1)<3. Согласно некоторым вариантам реализации изобретения, 3' концы фрагментов отличаются по сравнению с последовательностью-мишенью на 1 основание; соответственно, согласно некоторым вариантам реализации изобретения, (у2-y1)=1.
Согласно некоторым вариантам реализации изобретения, уникальный индекс («маркер» согласно некоторым вариантам реализации изобретения) используется для связывания фрагмента с матрицей нуклеиновой кислоты, из которой он был получен. Согласно некоторым вариантам реализации изобретения, уникальный индекс представляет собой синтетические нуклеотиды с уникальной последовательностью или природное нуклеотиды с уникальной последовательностью, которые позволяют легко идентифицировать нуклеиновую кислоту-мишень в пределах сложной группы олигонуклеотидов (например, фрагментов), содержащих различные последовательности. Согласно конкретным вариантам реализации изобретения, уникальные индексные идентификаторы присоединяют к фрагментам нуклеиновой кислоты до присоединения адаптерных последовательностей. Согласно некоторым вариантам реализации изобретения, уникальные индексные идентификаторы содержатся в пределах адаптерных последовательностей таким образом, что уникальная последовательность содержится в прочтениях секвенирования. Эти данные подтверждают, что гомологичные фрагменты можно выявлять на основе уникальных индексов, которые присоединены к каждому фрагменту, обеспечивая, таким образом, также точную реконструкцию консенсусной последовательности. Гомологичные фрагменты могут возникать, например, случайным образом из-за геномных повторов, двух фрагментов, происходящих их гомологичных хромосом, или фрагментов, происходящих из перекрывающихся участков на одной хромосоме. Гомологичные фрагменты также могут возникать из близко родственных последовательностей (например, близко родственных членов семейства генов, паралогов, ортологов, онологов, ксенологов и/или псевдогенов). Такие фрагменты могут быть удалены для подтверждения того, что сборка длинных фрагментов может быть однозначно рассчитана. Маркеры можно присоединять, как описано выше для адаптерных последовательностей. Индексы (например, маркеры) могут быть включены в последовательности адаптеров.
Согласно некоторым вариантам реализации изобретения, уникальный индекс (например, индексный идентификатор, метку, маркер и т.д.) представляет собой «штрихкод». При использовании в настоящей заявке, термин «штрихкод» относится к известной последовательности нуклеиновой кислоты, которая обеспечивает некоторые признаки нуклеиновой кислоты, с которой связан штрихкод связан для возможности идентификации. Согласно некоторым вариантам реализации изобретения, признак нуклеиновой кислоты, которую предполагается идентифицировать, представляет собой образец или источник, из которого получена нуклеиновая кислота. Согласно некоторым вариантам реализации изобретения, штрихкоды составляют по меньшей мере 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 или более нуклеотидов в длину. Согласно некоторым вариантам реализации изобретения, штрихкоды короче чем 10, 9, 8, 7, 6, 5 или 4 нуклеотидов в длину. Согласно некоторым вариантам реализации изобретения, длина штрихкодов, связанных с некоторыми нуклеиновыми кислотами, отличается от длины штрихкодов, связанных с другими нуклеиновыми кислотами. В целом, штрихкоды имеют достаточную длину и включают последовательности, которые достаточно различны для возможности идентификации образцов на основе штрихкодов, с которыми они связаны. Согласно некоторым вариантам реализации изобретения, штрихкод и источник образца, с которым он связан, можно точно идентифицировать после возникновения мутации, инсерции или делеции одного или более нуклеотидов в штрихкод-последовательности, например, мутация, инсерция или делеция 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более нуклеотидов. Согласно некоторым вариантам реализации изобретения, каждый штрихкод из множества штрихкодов отличается от каждого другого штрихкода из указанного множества по двум или более положениям нуклеотидов, например, по 2, 3, 4, 5, 6, 7, 8, 9, 10 или более положениям. Согласно некоторым вариантам реализации изобретения, один или более адаптеров включают (включает) по меньшей мере одну из множества последовательностей штрихкода. Согласно некоторым вариантам реализации изобретения, способы согласно технологии дополнительно включают идентификацию образца или источника, из которого произошла нуклеиновая кислота-мишень, на основе штрихкод-последовательности, с которой соединена указанная нуклеиновая кислота-мишень. Согласно некоторым вариантам реализации изобретения, способы согласно технологии дополнительно включают идентификацию нуклеиновой кислоты- мишени на основе штрихкод-последовательности, с которой соединена указанная нуклеиновая кислота-мишень. Некоторые варианты реализации способов дополнительно включают идентификацию источника или образца нуклеотидной последовательности-мишени путем определения нуклеотидной последовательности-штрихкод. Некоторые варианты реализации способов дополнительно включают приложения для подсчета молекул (например, цифровой нумерации штрихкода и/или группировки) для определения уровня экспрессии или количества копий желаемых мишеней. В целом, штрихкод может включать последовательность нуклеиновой кислоты, которая при связывании с нуклеиновой кислотой-мишенью служит в качестве идентификатора образца, из которого был получен полинуклеотид-мишень.
Согласно некоторым вариантам реализации изобретения, способы обеспечивают последовательность, содержащую до 100 оснований или, согласно некоторым вариантам реализации изобретения, более чем 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000 или более оснований. Согласно некоторым вариантам реализации изобретения, технология обеспечивает последовательность, содержащую более чем 1000 оснований, например, более чем 2000, 2500, 3000, 3500, 4000, 4500 или 5000 или более оснований. Согласно некоторым вариантам реализации изобретения, консенсусная последовательность содержит до 100 оснований или более, например, 200, 300, 400, 500, 600, 700, 800, 900, 1000 или более оснований; согласно некоторым вариантам реализации изобретения, консенсусная последовательность содержит более чем 1000 оснований, например, более чем 2000, 2500, 3000, 3500, 4000, 4500 или 5000 или более оснований.
Согласно некоторым вариантам реализации изобретения, олигонуклеотид, такой как праймер, адаптер и т.д., содержит «универсальную» последовательность. Универсальная последовательность представляет собой известную последовательность, например, для применения в качестве сайта связывания праймера или зонда с использованием праймера или зонда с известной последовательностью (например, комплементарной универсальной последовательности). Тогда как специфичная по отношению к матрице последовательность праймера, последовательность праймера со штрихкодом и/или последовательность адаптера со штрихкодом может различаться согласно вариантам реализации технологии, например, между фрагментами, между образцами, между источниками или между интересующими областями, согласно вариантам реализации технологии, универсальная последовательность является одинаковой среди фрагментов, образцов, источников или интересующих областей таким образом, что со всеми фрагментами, содержащими универсальную последовательность, можно проводить манипуляции и/или обрабатывать их одинаковым или сходным образом, например, амплифицирвоать, идентифицировать, секвенировать, выделять и т.д., с использованием сходных способов или технологий (например, с использованием одинаковых праймеров или зондов).
Согласно конкретным вариантам реализации изобретения, используется праймер, содержащий универсальную последовательность (например, универсальную последовательность А), штрихкод-последовательность и специфичную по отношению к матрице последовательность. Согласно конкретным вариантам реализации изобретения, используется первый адаптер, содержащий универсальную последовательность (например, универсальную последовательность В), и согласно конкретным вариантам реализации изобретения, используется второй адаптер, содержащий универсальную последовательность (например, универсальную последовательность С). Универсальная последовательность А, универсальная последовательность В и универсальная последовательность С могут представлять собой любую последовательность. Указанная номенклатура используется для того, чтобы показать, что универсальная последовательность А первой нуклеиновой кислоты (например, фрагмента), содержащей универсальную последовательность А, является такой же, как универсальная последовательность А второй нуклеиновой кислоты (например, фрагмента), содержащей универсальную последовательность А, универсальная последовательность В первой нуклеиновой кислоты (например, фрагмента), содержащей универсальную последовательность В, является такой же, как универсальная последовательность В второй нуклеиновой кислоты (например, фрагмента), содержащей универсальную последовательность В, и универсальная последовательность С первой нуклеиновой кислоты (например, фрагмента), содержащей универсальную последовательность С, является такой же, как универсальная последовательность С второй нуклеиновой кислоты (например, фрагмента), содержащей универсальную последовательность С. Тогда как универсальные последовательности А, В и С в целом различаются согласно вариантам реализации технологии, они не обязательно являются различными. Таким образом, согласно некоторым вариантам реализации изобретения, универсальные последовательности А и В являются одинаковыми; согласно некоторым вариантам реализации изобретения, универсальные последовательности В и С являются одинаковыми; согласно некоторым вариантам реализации изобретения, универсальные последовательности А и С являются одинаковыми; и согласно некоторым вариантам реализации изобретения, универсальные последовательности А, В и С являются одинаковыми. Согласно некоторым вариантам реализации изобретения, универсальные последовательности А, В и С являются разными.
Например, если предполагается секвенировать две интересующие области (например, из одного или разных источников или, например, из двух разных областей одной нуклеиновой кислоты, хромосомы, гена и т.д.), могут использоваться два праймера, где один праймер содержит первую специфичную в отношении матрицы последовательность для праймирования с первого интересующего участка и первый штрихкод для связывания первого амплифицированного продукта с первым интересующим участком, и второй праймер содержит вторую специфичную в отношении матрицы последовательность для праймирования со второго интересующего участка и второй штрихкод для связывания второго амплифицированного продукта со второй интересующей областью. Однако указанные два праймера, согласно некоторым вариантам реализации изобретения, включают одну и ту же универсальную последовательность (например, универсальную последовательность А) для получения пула и последующей совместной обработки. Могут использоваться две или более универсальных последовательностей. В целом, число универсальных последовательностей будет меньше, чем число специфичных по отношению к мишени последовательностей и/или штрихкод-последовательностей для получения пула образцов и обработки пулов как одного образца (партии).
Соответственно, согласно некоторым вариантам реализации изобретения, определение первой нуклеотидной субпоследовательности и второй нуклеотидной субпоследовательности включает праймирование универсальной последовательностью. Согласно некоторым вариантам реализации изобретения, определение первой нуклеотидной субпоследовательности и второй нуклеотидной субпоследовательности включает терминацию полимеризации с помощью содержащего 3'-О-блокирующую группу нуклеотидного аналога. Например, согласно некоторым вариантам реализации изобретения, определение первой нуклеотидной субпоследовательности и второй нуклеотидной субпоследовательности включает терминацию полимеризации с помощью 3'-О-алкинил-нуклеотидного аналога, например, согласно некоторым вариантам реализации изобретения, определение первой нуклеотидной субпоследовательности и второй нуклеотидной субпоследовательности включает терминацию полимеризации с помощью 3'-О пропаргил-нуклеотидного аналога. Согласно некоторым вариантам реализации изобретения, определение первой нуклеотидной субпоследовательности и второй нуклеотидной субпоследовательности включает терминацию полимеризации с помощью нуклеотидного аналога, содержащего обратимый терминатор.
Полученные короткие прочтения последовательности разделяют согласно их штрихкоду (например, извлекают в отдельные файлы), и прочтения, происходящие из одних и тех же образцов, источников, интересующих областей и т.д. группируют вместе, например, сохраняют в отдельных файлах или помещают в организованные структуры данных, позволяющие группировать прочтения, подлежащие идентификации соответствующим образом. Затем сгруппированные короткие последовательностей собираются в консенсусные последовательности. Сборку последовательностей в целом можно разделить на две большие категории: сборка de novo и сборка картированием референсного генома. При сборке de novo прочтения последовательности собираются вместе таким образом, что они образуют новую и ранее не известную последовательность. При картировании к референсному геному прочтения последовательности собираются на основании существующего скелета последовательности (например, референсной последовательности и т.д.) для построения последовательности, которая подобна, но не обязательно идентична указанному скелету последовательности.
Таким образом, согласно некоторым вариантам реализации изобретения, нуклеиновые кислоты-мишени, соответствующие каждой интересующей области, реконструируют с помощью сборки de-novo. Для начала процесса реконструкции короткие прочтения сшивают вместе с помощью биоинформационного способа поиска перекрытий и их удлинения с получением консенсусной последовательности. Согласно некоторым вариантам реализации изобретения, способ дополнительно включает картирование консенсусной последовательности к референсной последовательности. Способы согласно технологии обладают преимуществом высокой балльной оценки качества секвенирования, которая обеспечивает надежное распознавание оснований для реконструкции полноразмерных фрагментов. Кроме сборки de-novo фрагменты можно использовать для получения информации фазирования (приписываемой гомологичным копиям хромосом) геномных вариантов путем наблюдения происхождения консенсусных последовательностей из одной из любых хромосом.
Согласно некоторым вариантам реализации изобретения, компьютерная система обеспечивается для сборки и биоинформационной обработки информации последовательности (например, идентификации штрихкодов, распределения, группировки, распознавание оснований, определения консенсусной идентичности каждого основание, сшивки прочтений, оценки баллов качества, выравнивания прочтений и/или консенсусных последовательностей к референсной последовательности и т.д.). Согласно некоторым вариантам реализации, компьютерная система включает шину или другой механизм связи для обмена информацией и процессор, соединенный с шиной для обработки информации. Согласно некоторым вариантам реализации, компьютерная система включает память, которая может представлять собой память с произвольной выборкой (RAM) или другое устройство динамической памяти, соединенное с шиной, и инструкции, подлежащие выполнению процессором. Память также может использоваться для хранения временных переменных или другой промежуточной информации во время осуществления инструкции, подлежащей выполнению процессором. Согласно некоторым вариантам реализации, компьютерная система дополнительно включает постоянное запоминающее устройство (РЗУ) или другое статичное накопительное устройство, сопряженное с шиной для хранения статичной информации и инструкции для процессора. Согласно некоторым вариантам реализации изобретения, накопительное устройство, такое как твердотельный диск (например, «флэш» память), магнитный диск или оптический диск, предоставлено и сопряжено с шиной для хранения информации и инструкций.
Согласно некоторым вариантам реализации, компьютерная система сопряжена через шину с монитором, таким как электронно-лучевая трубка (CRT) или жидкокристаллический дисплей (LCD), для отображения информации для пользователя компьютера. Согласно некоторым вариантам реализации изобретения, входное устройство, включая буквенно-цифровые и другие клавиши, сопряжено с шиной для обмена информацией и выбора команд для процессора. Другой тип устройства пользовательского ввода представляет собой управление курсором, такое как мышь, следящий точечный курсор или клавиши управления курсором для сообщения директивной информации и выбора команд для процессора и для контроля движений курсора на экране.
Согласно некоторым вариантам реализации изобретения, компьютерная система осуществляет аспекты технологии согласно настоящему изобретению. В соответствии с конкретными вариантами реализации технологии, результаты предоставляются компьютерной системой в ответ на выполнение процессором одной или более последовательностей одной или более инструкций, содержащихся в памяти. Такие инструкции могут считываться в память из другого машиночитаемого носителя, такого как накопительное устройство. Альтернативно, жесткая схема может использоваться вместо или в комбинации с инструктирующими программами для осуществления технологии согласно настоящему изобретению. Таким образом, реализация идей согласно настоящему изобретению не ограничивается какой-либо конкретной комбинацией жесткой схемы и программного обеспечения. Например, как описано в настоящей заявке, варианты реализации технологии включают применение, хранение и передачу данных с использованием «облачной» компьютерной технологии, проводной (такой как оптоволокно, кабель, медь, ADSL, Ethernet и т.п.) и/или беспроводной технологии (например, IEEE 802,11 и т.п.). Как описано в настоящей заявке, согласно некоторым вариантам реализации изобретения, компоненты технологии связаны через локальную вычислительную сеть (LAN), беспроводную локальную вычислительную сеть (WLAN), глобальную вычислительную сеть (WAN), такую как интернет, или сеть любого другого типа, топологии и/или протокола. Согласно некоторым вариантам реализации изобретения, технология включает применение переносного устройства, такого как портативный компьютер, смартфон, планшетный компьютер, ноутбук, карманный компьютер, компьютер Hiptop, например, для воспроизведения результатов, принятия данных от пользователя, предоставление инструкций для другого компьютера, хранения данных и/или осуществления других этапов способов, предложенных в настоящей заявке. Согласно некоторым вариантам реализации изобретения, предложено применение «тонкого» клиентского терминала для воспроизведения результатов, принятия входных данных от пользователя, обеспечения инструкций для другого компьютера, хранения данных и/или осуществления других этапов способов, предложенных в настоящей заявке.
Согласно некоторым вариантам реализации изобретения, предложен способ определения нуклеотидной последовательности-мишени, включающий определение n нуклеотидных субпоследовательностей нуклеотидной последовательности-мишени (обозначенной буквой m), где 5' конец нуклеотидной субпоследовательности m находится на нуклеотиде xm нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде ym нуклеотидной последовательности-мишени; 5' конец нуклеотидной субпоследовательности (m+1) находится на нуклеотиде xm+1 нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде ym+1 нуклеотидной последовательности-мишени; и сборку n нуклеотидных субпоследовательностей для получения консенсусной последовательности для нуклеотидной последовательности-мишени, где m варьирует от 1 до n; xm+1<ym; и (ym-xm)<100, 90, 80, 70, 60, 50, 55, 50, 45, 40, 35 или 30 или less, (ym+1-xm+1)<100, 90, 80, 70, 60, 50, 55, 50, 45, 40, 35 или 30 или менее, и (ym+1-ym)<20, 10 или менее или менее чем 5, 4 или 3 или равен 1. Согласно некоторым вариантам реализации изобретения, фрагменты содержат менее чем 50 п.о.; соответственно, согласно некоторым вариантам реализации изобретения, (ym-xm)<50, и (ym+1-xm+1)<50. Согласно некоторым вариантам реализации изобретения, фрагменты содержат менее чем 40 п.о.; соответственно, согласно некоторым вариантам реализации изобретения, (ym-xm)<40, и (ym+1-xm+1)<40. Согласно некоторым вариантам реализации изобретения, фрагменты содержат менее чем 30 п.о.; соответственно, согласно некоторым вариантам реализации изобретения, (ym-xm)<30, и (ym+1-xm+1)<30.
Согласно некоторым вариантам реализации изобретения, 3' концы фрагментов различаются на 4 или 3 основания по сравнению с последовательностью нуклеиновой кислоты-мишени. Соответственно, согласно некоторым вариантам реализации изобретения, (ym+1-ym)<4, или (ym+1-ym)<3. Согласно некоторым вариантам реализации изобретения, 3' концы фрагментов различаются на 1 основание по сравнению с последовательностью нуклеиновой кислоты-мишени. Таким образом, согласно некоторым вариантам реализации изобретения, (ym+1-ym)=1.
Согласно некоторым вариантам реализации изобретения, определение n нуклеотидных субпоследовательностей включает праймирование универсальной последовательностью. Согласно некоторым вариантам реализации изобретения, определение n нуклеотидных субпоследовательностей включает терминацию полимеризации с помощью содержащего 3'-О-блокирующую группу нуклеотидного аналога. Согласно некоторым вариантам реализации изобретения, определение первой нуклеотидной субпоследовательности и второй нуклеотидной субпоследовательности включает терминацию полимеризации с помощью 3'-О-алкинил-нуклеотидного аналога. Согласно некоторым вариантам реализации изобретения, определение первой нуклеотидной субпоследовательности и второй нуклеотидной субпоследовательности включает терминацию полимеризации с помощью 3'-О-пропаргил-нуклеотидного аналога. Согласно некоторым вариантам реализации изобретения, определение первой нуклеотидной субпоследовательности и второй нуклеотидной субпоследовательности включает терминацию полимеризации с помощью нуклеотидного аналога, содержащего обратимый терминатор.
Согласно некоторым вариантам реализации изобретения, предложены способы создания библиотеки для секвенирования нового поколения. Согласно некоторым вариантам реализации изобретения, способы включают амплификацию нуклеотидной последовательности-мишени с использованием праймера, содержащего специфичную по отношению к мишени последовательность, универсальную последовательность А и нуклеотидную последовательность-штрихкод, связанную с нуклеиновой кислотой-мишенью, для получения поддающегося идентификации ампликона; лигирование первого адаптерного олигонуклеотида, содержащего универсальную последовательность В, с 3' концом ампликона для образования комплекса адаптер-ампликон; циркуляризацию комплекса адаптер-ампликон для образования кольцевой матрицы; создание «лестницы» библиотеки фрагментов на основе кольцевой матрицы с использованием содержащего 3'-О-блокирующую группу нуклеотидного аналога и лигирование второго адаптерного олигонуклеотида, содержащего универсальную последовательность С, с 3' концами фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для создания библиотеки для секвенирования нового поколения (например, с использованием лигазы или химического лигировани, например, с помощью реакии «клик-химии», например, катализируемой медью реакции алкина (например, 3' алкина) и азида (например, 5' азида)).
Согласно некоторым вариантам реализации изобретения, нуклеотидная последовательность-штрихкод содержит 1 до 20 нуклеотидов. Согласно некоторым вариантам реализации изобретения, первый адаптерный олигонуклеотид содержит от 10 до 80 нуклеотидов. Согласно некоторым вариантам реализации изобретения, нуклеотидные последовательности фрагментов библиотеки, представляющей собой «лестницу» фрагментов, соответствуют перекрывающимся нуклеотидным субпоследовательностям в пределах нуклеотидной последовательности-мишени, и 3' концы нуклеотидных последовательностей фрагментов соответствуют разным нуклеотидам нуклеотидной последовательности-мишени. Согласно некоторым вариантам реализации изобретения, нуклеотидные последовательности фрагментов библиотеки, представляющей собой «лестницу» фрагментов, включают менее чем 100 нуклеотидов, например, менее чем 90, 80, 70, 60, 50 или 40 нуклеотидов, например, от 15 до 50, например, от 15 до 40 нуклеотидов.
Согласно некоторым вариантам реализации изобретения, первый адаптерный олигонуклеотид содержит одноцепочечную ДНК, и/или второй адаптерный олигонуклеотид содержит одноцепочечную ДНК.
Согласно некоторым вариантам реализации изобретения, создание библиотеки, представляющей собой «лестницу» фрагментов, включает использование олигонуклеотидного праймера, комплементарного универсальной последовательности А.
Согласно некоторым вариантам реализации изобретения, способы дополнительно включают амплификацию библиотеки для секвенирования нового поколения.
Согласно некоторым вариантам реализации изобретения, 3'-О-алкинил-нуклеотидный аналог представляет собой 3'-О-пропаргил-нуклеотидный аналог. Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог содержит обратимый терминатор.
Технология дополнительно обеспечивает способы определения последовательности нуклеиновой кислоты. Например, согласно некоторым вариантам реализации изобретения, способ включает создание библиотеки для секвенирования нового поколения в соответсвии с технологией, предложенной в настоящей заявке; определение нуклеотидной последовательности фрагмента библиотеки, представляющей собой «лестницу» фрагментов, где указанная нуклеотидная последовательность содержит нуклеотидную субпоследовательность нуклеотидной последовательности-мишени; и определение нуклеотидной последовательности-штрихкода фрагмента библиотеки, представляющей собой «лестницу» фрагментов.
Согласно некоторым вариантам реализации изобретения, определение нуклеотидной последовательности фрагмента библиотеки, представляющей собой «лестницу» фрагментов, включает использование олигонуклеотидного праймера, комплементарного универсальной последовательности С. Кроме того, согласно некоторым вариантам реализации изобретения, определение нуклеотидной последовательности-штрихкода фрагмента библиотеки, представляющей собой «лестницу» фрагментов, включает использование олигонуклеотидного праймера, комплементарного универсальной последовательности В.
Согласно некоторым вариантам реализации изобретения, нуклеотидная последовательность фрагмента библиотеки, представляющей собой «лестницу» фрагментов, содержит менее чем 100 нуклеотидов, например, от 15 до 50 нуклеотидов, например, от 20 до 50, например, от 25 до 50, например, от 30 до 50, например, от 35 до 50, например, от 40 до 50 нуклеотидов. Согласно некоторым вариантам реализации изобретения, способы дополнительно включают объединение нуклеотидной последовательности-штрихкода с источником нуклеотидной последовательности-мишени.
Согласно некоторым вариантам реализации изобретения, способы дополнительно включают сбор или группировку нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, имеющих одинаковую нуклеотидную последовательность-штрихкод. Согласно некоторым вариантам реализации изобретения, способы дополнительно включают сборку множества нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для получения консенсусной последовательности. Согласно некоторым вариантам реализации изобретения, способы дополнительно включают картирование консенсусной последовательности к референсной последовательности.
Согласно некоторым вариантам реализации изобретения, для реконструкции консенсусной последовательности технология включает присоединение меток к нуклеиновым кислотам, таких как связывающие нуклеиновые кислоты белки, оптические метки, нуклеотидные аналоги и другие метки, известные в данной области техники.
Технология обеспечивает связанные композиции, содержащие библиотеки для секвенирования нового поколения, где указанные библиотеки для секвенирования нового поколения содержат множество нуклеиновых кислот, где каждая нуклеиновая кислота содержит универсальную последовательность А, нуклеотидную последовательность-штрихкод, вторую универсальную последовательность В, нуклеотидную субпоследовательность нуклеотидной последовательности-мишени и универсальную последовательность С. Согласно некоторым вариантам реализации изобретения, композиции содержат n нуклеиновых кислот, где 5' конец нуклеотидной субпоследовательности m находится на нуклеотиде xm нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде ym нуклеотидной последовательности-мишени; 5' конец нуклеотидной субпоследовательности (m+1) находится на нуклеотиде xm+1 нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде ym+1 нуклеотидной последовательности-мишени; m варьирует от 1 до n; xm=xm+1; и (ym+1-ym)<20, 10 или менее чем 5, 4, 3 или 2. Согласно некоторым вариантам реализации изобретения, 3' концы фрагментов библиотеки для секвенирования смещены по отношению друг к другу и нуклеотидной последовательности-мишени на 4 или 3 основания; соответственно, согласно некоторым вариантам реализации изобретения, (ym+1-ym)<4 или (ym+1-ym)<3. Согласно некоторым вариантам реализации изобретения, 3' концы фрагментов библиотеки для секвенирования смещены по отношению друг к другу и нуклеотидной последовательности-мишени на 1 основание; соответственно, согласно некоторым вариантам реализации изобретения, (ym+1-ym)=1.
Согласно некоторым вариантам реализации изобретения, универсальная последовательность В содержит от 10 до 100 нуклеотидов, и/или нуклеотидная последовательность-штрихкод содержит от 1 до 20 нуклеотидов.
Согласно некоторым вариантам реализации изобретения, композиции дополнительно включают 3'-О-блокированный нуклеотидный аналог, такой как 3'-О-алкинил-нуклеотидный аналог, например, 3'-О-пропаргил-нуклеотидный аналог. Согласно некоторым вариантам реализации изобретения, композиции дополнительно содержат праймер для секвенирования. Например, согласно некоторым вариантам реализации изобретения, композиции дополнительно содержат праймер для секвенирования, комплементарный универсальной последовательности С, и/или праймер для секвенирования, комплементарный универсальной последовательности В.
Согласно некоторым вариантам реализации изобретения, нуклеотидная последовательность-штрихкод связана с нуклеотидной последовательностью-мишенью. Согласно некоторым вариантам реализации изобретения, множество нуклеиновых кислот содержит нуклеиновые кислоты, имеющие разные нуклеотидные последовательности-штрихкоды и разные нуклеотидные субпоследовательности нуклеотидной последовательности-мишени, где каждая нуклеотидная последовательность-штрихкод связана с нуклеотидной последовательностью-мишенью. Согласно некоторым вариантам реализации изобретения, нуклеотидная последовательность-штрихкод связана с абсолютным соответствием с нуклеотидной последовательностью-мишенью.
Согласно некоторым вариантам реализации изобретения, каждая нуклеиновая кислота библиотеки для секвенирования нового поколения содержит 3'-О-блокированный нуклеотидный аналог, например, 3'-Оалкинил-нуклеотидный аналог, например, 3'-О-пропаргил-нуклеотидный аналог. Согласно некоторым вариантам реализации изобретения, каждая нуклеиновая кислота библиотеки для секвенирования нового поколения содержит нуклеотидный аналог, содержащий обратимый терминатор.
Также предложены наборы для получения библиотеки для секвенирования NGS и/или для получения информации последовательности на основе нуклеиновой кислоты-мишени. Согласно некоторым вариантам реализации технологии, предложен набор, содержащий нуклеотидный аналог, например, для получения «лестницы» нуклеотидных фрагментов в соответствии со способами, предложенными в настоящей заявке. Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог представляет собой 3'-О-блокированный нуклеотидный аналог, например, 3'-О-алкинил-нуклеотидный аналог, например, 3'-О-пропаргил-нуклеотидный аналог. Согласно некоторым вариантам реализации изобретения, в наборе предложены стандартные нуклеотиды А, С, G, U и/или Т, а также один или более (например, 1, 2, 3 или 4) А, С, G, U и/или Τ нуклеотидных аналогов.
Согласно некоторым вариантам реализации изобретения, наборы содержат полимеразу (например, природную полимеразу, модифицированную полимеразу и/или сконструированную полимеразу и т.д.), например, для амплификации (например, путем термоциклирования, изотермической амплификации) или для секвенирования и т.д. Согласно некоторым вариантам реализации изобретения, наборы включают лигазу, например, для присоединения адаптеров к нуклеиновой кислоте, такой как ампликон или фрагмент «лестницы», или для циркуляризации комплекса адаптер-ампликон. Некоторые варианты реализации наборов включают катализаторы-реагенты на основе меди, например, для реакции «клик-химии», например, для проведения реакции азида и алкинильной группы для образования триазольной связи. Некоторые варианты реализации наборов обеспечивают буферы, соли, реакционные сосуды, инструкции и/или компьютерные программы.
Согласно некоторым вариантам реализации изобретения, наборы включают праймеры и/или адаптеры. Согласно некоторым вариантам реализации изобретения, адаптеры включают химическую модификацию, подходящую для присоединения адаптера к нуклеотидному аналогу, например, с помощью реакций «клик-химии». Например, согласно некоторым вариантам реализации изобретения, набор содержит нуклеотидный аналог, содержащий алкиновую группу и адаптерный олигонуклеотид, содержащий азидную группу (N3). Согласно некоторым вариантам реализации изобретения, процесс «клик-химии», такой как азид-алкиновое циклоприсоединение, используется для связывания адаптера с фрагментом через образование триазола.
Некоторые варианты реализации технологии обеспечивают системы для получения информации последовательности. Например, варианты реализации системы включают нуклеотидный аналог для получения «лестницы» фрагментов из нуклеиновой кислоты-мишени и расположенную на машиночитаемом носителе инструкцию для определения последовательности нуклеиновой кислоты-мишени на основе сборки коротких прочтений последовательности. Согласно некоторым вариантам реализации изобретения, системы включают один или более адаптерных олигонуклеотидов (например, подходящих для присоединения к нуклеотидным аналогам) или другим компонентам набора, как описано выше.
Например, некоторые варианты реализации системы связаны со сборкой (сшиванием, реконструированием) последовательности нуклеиновой кислоты. Варианты реализации таких систем включают различные компоненты, такие как, например, секвенатор нуклеиновых кислот, хранение данных образца последовательности, накопитель данных референсной последовательности и аналитическое вычислительное устройство/сервер/узел. Согласно некоторым вариантам реализации изобретения, аналитическое вычислительное устройство/сервер/узел представляет собой рабочую станцию, универсальный компьютер, персональный компьютер, мобильное устройство и т.д. Согласно некоторым вариантам реализации изобретения, системы включают функциональные средства для идентификации штрихкода, анализа последовательностей на основе штрихкода и группировку последовательностей, имеющих общие штрихкоды.
Согласно некоторым вариантам реализации изобретения, секвенатор нуклеиновых кислот настроен на анализ (например, распознавание) фрагмента нуклеиновой кислоты (например, одного фрагмента, фрагментов-партнеров, фрагмента со спаренными концами и т.д.) с использованием всего доступного разнообразия способов, платформ или технологий для получения информации последовательности нуклеиновой кислоты. Согласно некоторым вариантам реализации изобретения, системы включают функциональные средства для обеспечения распознавания основания, балльной оценки качества, выравнивания последовательностей, идентификации штрихкода, анализа последовательностей на основе штрихкода и группировки последовательностей, имеющих общие штрихкоды.
Согласно некоторым вариантам реализации, секвенатор нуклеиновой кислоты сообщается с накопителем данных последовательности образца либо непосредственно через кабель данных (например, последовательный кабель, прямое кабельное соединение и т.д.) или шинное соединение или, альтернативно, через сетевое соединение (например, интернет, LAN, WAN, WLAN, VPN и т.д.). Согласно некоторым вариантам реализации, сетевое соединение представляет собой жесткое механическое соединение. Например, согласно некоторым вариантам реализации изобретения, предложен секвенатор нуклеиновых кислот, коммуникативно присоединенный (через Category 5 (САТ5), оптоволокно или эквивалентную кабельную сеть) к серверу данных, который, в свою очередь, коммуникативно связан (через САТ5, оптоволокно или эквивалентную кабельную сеть) через интернет с накопителем данных образца последовательности. Согласно некоторым вариантам реализации, сетевое соединение представляет собой беспроводное сетевое соединение (например, Wi-Fi, WLAN и т.д.), например, использующее IEEE 802,11 (например, a/b/g/n и т.д.) или эквивалентный формат передачи данных. На практике используемое сетевое соединение зависит от конкретных требований системы. Согласно некоторым вариантам реализации, накопитель данных образца последовательности представляет собой интегрированный компонент секвенатора нуклеиновых кислот.
Согласно некоторым вариантам реализации изобретения, накопитель данных образца последовательности представляет собой устройство хранения базы данных, систему или обеспечение (например, раздел накопителя данных и т.д.), настроенное на организацию и хранение данных прочтения последовательности нуклеиновой кислоты, генерированных секвенатором нуклеиновых кислот (например, коротких перекрывающихся прочтений последовательности, составляющих менее чем 300 или менее чем 200 оснований, например, ~30-50 оснований) и связанной индексной информации, такой как последовательность - штрихкод и метаинформация, связанная со штрихкодом, такая как источник и тип образца, нуклеиновая кислота-мишень, интересующая область, экспериментальные условия, клинические данные и т.д.) таким образом, что данные можно искать (например, с помощью последовательности-штрихкод или связанной метаинформации) и извлекать вручную (например, с помощью администратора/оператора-клиента базы данных) или автоматически с помощью компьютерной программы/приложения/программного сценария. Согласно некоторым вариантам реализации, накопитель референсных данных может представлять собой любое устройство базы данных, накопительную систему или обеспечение (например, раздел накопителя данных и т.д.), настроенное на организацию и хранение референсных последовательностей (например, полного/части генома, полного/части экзома, гена, области, хромосомы, ВАС и т.д.) таким образом, что данные можно искать и извлекать вручную (например, с помощью администратора/оператора-клиента базы данных) или автоматически с помощью компьютерной программы/приложения/программного сценария. Согласно некоторым вариантам реализации, данные прочтения образца секвенирования нуклеиновой кислоты хранятся на накопителе данных образца последовательности и/или накопителе референсных данных в файлах различных типов/форматов разных данных, включая, но не ограничиваясь указанными: *.fasta, *.csfasta, *seq.txt, *qseq.txt, *.fastq, *.sff, *prb.txt, *.sms, *srs и/или *.qv.
Согласно некоторым вариантам реализации изобретения, накопитель данных образца последовательности и накопитель референсных данных независимо представляют собой автономные устройства/системы или реализуются на разных устройствах. Согласно некоторым вариантам реализации изобретения, накопитель данных образца последовательности и накопитель референсных данных реализуют на одном и том же устройстве/система. Согласно некоторым вариантам реализации изобретения, накопитель данных образца последовательности и/или накопитель референсных данных обеспечиваются на аналитическом вычислительном устройстве/сервере/узле.
Согласно некоторым вариантам реализации изобретения, аналитическое вычислительное устройство/сервер/узел сообщается с накопителем данных образца последовательности и накопителем референсных данных либо прямо через кабель данных (например, последовательный кабель, прямое кабельное соединение и т.д.) или шинное соединение, либо, альтернативно, через сетевое соединение (например, интернет, LAN, WAN, VPN и т.д.). Согласно некоторым вариантам реализации, аналитическое вычислительное устройство/сервер/узел расположен на ассемблере, например, механизме референсного картирования или модуле de novo картирования и/или механизме третичного анализа.
Согласно некоторым вариантам реализации изобретения, модуль картирования de novo настраивают для сборки прочтений образца последовательности нуклеиновой кислоты с накопителя данных образца в новые и ранее не известные последовательности.
Согласно некоторым вариантам реализации изобретения, механизм референсного картирования настраивают для получения прочтений образца последовательности нуклеиновой кислоты (например, которые имеют общий штрихкод и были сгруппированы вместе) из накопителя данных образца и картирования их по отношению к одной или более референсным последовательностям, полученным из накопителя референсных данных для сборки прочтений в последовательность, которая подобна, но не обязательно идентична референсной последовательности, с использованием всего разнообразия техник и способов референсного картирования/выравнивания. Заново собранную последовательность затем можно дополнительно анализировать с помощью одного или более необязательных механизмов третичного анализа для идентификации различий в генетическом строении (генотипе, гаплотипе), экспрессии гена или эпигенетическом статусе индивида, которые могут приводить к большим различиям в физических характеристиках (фенотипе). Например, согласно некоторым вариантам реализации, механизм третичного анализа настроен для идентификации различных геномных вариантов (в собранной последовательности), вызванных мутациями, рекомбинацией/кроссинговером или дрейфом генов; для идентификации фазирования генетической информации; для идентификации филогенетической и/или таксономической информации; для идентификации индивидов; для идентификации вида, рода или другой филогенетической классификации; для идентификации маркера лекарственной устойчивости или маркера восприимчивости к лекарственному средству (чувствительности); для идентификации слияния генов; для идентификации вариации числа копий; для идентификации статуса метилирования; для установления связи последовательности с заболеванием и т.д. Примеры типов геномных вариантов включают, но не ограничиваются указанными: однонуклеотидные полиморфизмы (SNP), вариации числа копий (CNV), инсерции/делеции («инсерционно-делеционные мутации»), инверсии, дупликации, транслокации, интеграции и т.д.
Необходимо понимать, однако, что различные механизмы и модули, размещенные на аналитическом вычислительном устройстве/сервере/узле, могут быть объединены или сжаты в один механизм или модуль в зависимости от требований конкретного приложения или архитектуры системы. Более того, согласно некоторым вариантам реализации, аналитическое вычислительное устройство/сервер/узел содержит дополнительные механизмы или модули согласно потребностям конкретного приложения или архитектуры системы.
Согласно некоторым вариантам реализации изобретения, механизмы картирования и/или третичного анализа настроены на обработку прочтений нуклеиновой кислоты и/или референсных последовательностей в цветовом пространстве. Согласно некоторым вариантам реализации, механизмы картирования и/или третичного анализа настроены на обработку прочтений нуклеиновой кислоты и/или референсных последовательности в базисном пространстве. Необходимо понимать, однако, что механизмы картирования и/или третичного анализа могут обрабатывать или анализировать данные последовательности нуклеиновой кислоты в любой схеме или формате, при условии что указанная схема или формат сохраняет идентичность оснований и положений последовательности нуклеиновой кислоты.
Согласно некоторым вариантам реализации изобретения, данные прочтения образца секвенирования нуклеиновой кислоты и референсной последовательности доставляются в аналитическое вычислительное устройство/сервер/узел в виде файлов различных типов/форматов разных входных данных, включая, но не ограничиваясь указанными: *.fasta, *.csfasta, *seq.txt, *qseq.txt, *.fastq, *.sff, *prb.txt, *.sms, *srs и/или *.qv.
Согласно некоторым вариантам реализации изобретения, предложен клиентский терминал. Клиентский терминал, согласно некоторым вариантам реализации изобретения, представляет собой «тонкое» или, согласно некоторым вариантам реализации изобретения, «толстое» клиентское вычислительное устройство. Согласно некоторым вариантам реализации изобретения, клиентский терминал включает интернет-браузер (например, Internet Explorer, Firefox, Safari, Chrome и т.д.), который используется для контроля работы механизма референсного картирования, модуля de novo картирования и/или механизма третичного анализа. Таким образом, клиентский терминал может иметь доступ к механизму референсного картирования, модулю de novo картирования и/или механизму третичного анализа с использованием браузера для контроля их функций. Например, клиентский терминал можно использовать для преобразования рабочих параметров (например, лимита ошибок, порогового значения оценки качества и т.д.) различных механизмов в зависимости от требований конкретного приложения. Подобным образом, клиентский терминал также может включать монитор для отображения результатов анализа, который осуществляется ассемблером, механизмом референсного картирования, модулем de novo картирования и/или механизмом третичного анализа.
Технология, предложенная в настоящей заявке, согласно вариантам реализации способа, композиции, набора и системы, применяется, например, для получения библиотеки NGS для секвенирования, для получения нуклеотидной последовательности, для картирования однонуклеотидного полиморфизма, для различения аллелей, для секвенирования генома, для идентификации редких минорных популяционных вариантов (например, соматических мутаций при раке или слабо распространенного патогена на фоне большого количества хозяйской или непатогенной ДНК) и т.д.
Секвенирование может представлять собой любой способ, известный в данной области техники. Согласно конкретным вариантам реализации изобретения, секвенирование представляет собой секвенирование путем синтеза. Согласно другим вариантам реализации, секвенирование представляет собой одномолекулярное секвенирование путем синтеза. Согласно конкретным вариантам реализации изобретения, секвенирование включает гибридизацию праймера с матрицей с получением дуплекса матрица/праймер, приведение в контакт указанного дуплекса с полимеразным ферментом в присутствии поддающихся выявлению меченых нуклеотидов в условиях, позволяющих зависимое от матрицы добавление нуклеотидов к праймеру с помощью полимеразы, выявление сигнала от включенного меченого нуклеотида и последовательное повторение этапов приведения в контакт и выявления по меньшей мере один раз, где последовательное выявление включенных меченых нуклеотидов обеспечивает определение последовательности нуклеиновой кислоты. Типичные поддающиеся выявлению метки включают радиоактивные метки, флуоресцентные метки, ферментативные метки и т.д. Согласно конкретным вариантам реализации изобретения, поддающаяся выявлению метка может представлять собой необязательно поддающуюся выявлению метку, такую как флуоресцентная метка. Типичные флуоресцентные метки (для секвенирования и/или для других целей, таких как мечение нуклеиновой кислоты, праймера, зонда и т.д.) включают цианин, родамин, флуоресцеин, кумарин, краситель BODIPY, Alexa или конъюгированные мультикрасители.
Согласно некоторым вариантам реализации изобретения, предложен способ для создания библиотеки для секвенирования нового поколения, включающий амплификацию нуклеотидной последовательностью-мишени с использованием праймера, содержащего специфичную по отношению к мишени последовательность, универсальную последовательность А и нуклеотидную последовательность-штрихкод (например, содержащую от 1 до 20 нуклеотидов), связанную с нуклеиновой кислотой- мишенью, для получения поддающегося идентификации ампликона; лигирование первого адаптерного олигонуклеотида (например, одноцепочечной ДНК, например, содержащей от 10 до 80 нуклеотидов), содержащего универсальную последовательность В, с 3' концом ампликона для образования комплекса адаптер-ампликон; циркуляризацию комплекса адаптер-ампликон с получением кольцевой матрицы; создание на основе указанной кольцевой матрицы с использованием праймера, комплементарного универсальной последовательности А, и содержащего 3'-О-блокирующую группу нуклеотидного аналога (например, 3'-О-алкинил-нуклеотидного аналога, 3'-О-пропаргил-нуклеотидного аналога или нуклеотидного аналога, содержащего обратимый терминатор) библиотеки, представляющей собой «лестницу» фрагментов, содержащей множество фрагментов; и лигирование (например, с помощью реакций «клик-химии», например, с использованием каталитического реагента на основе меди, например, для образования триазола из азида и алкинила) второго адаптерного олигонуклеотида (например, одноцепочечной ДНК), содержащего универсальную последовательность С, с 3' концами фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для создания библиотеки для секвенирования нового поколения, где нуклеотидные последовательности фрагментов библиотеки, представляющей собой «лестницу» фрагментов, содержат от 15 до 40 нуклеотидов и соответствуют перекрывающимся нуклеотидным субпоследовательностям в пределах нуклеотидной последовательности-мишени, и 3' концы указанных нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, соответствуют разным нуклеотидам нуклеотидной последовательности-мишени.
Согласно некоторым вариантам реализации изобретения, предложен способ определения нуклеотидной последовательности-мишени, включающий амплификацию нуклеотидной последовательности-мишени с использованием праймера, содержащего специфичную по отношению к мишени последовательность, универсальную последовательность А и нуклеотидную последовательность-штрихкод (например, содержащую от 1 до 20 нуклеотидов), связанную с нуклеиновой кислотой-мишенью, для получения ампликона; лигирование первого адаптерного олигонуклеотида (например, одноцепочечной ДНК, например, содержащей от 10 до 80 нуклеотидов), содержащего универсальную последовательность В, с 3' концом ампликона для образования комплекса адаптер-ампликон; циркуляризацию комплекса адаптер-ампликон для образования кольцевой матрицы; создание из указанной кольцевой матрицы с использованием праймера, комплементарного универсальной последовательности А, и содержащего 3'-О-блокирующую группу нуклеотидного аналога (например, 3'-О-алкинил-нуклеотидного аналога, 3'-О-пропаргил-нуклеотидного аналога или нуклеотидного аналога, содержащего обратимый терминатор) библиотеки, представляющей собой «лестницу» фрагментов, содержащей множество фрагментов; лигирование (например, с помощью реакций «клик-химии», например, с использованием каталитического реагента на основе меди, например, для образования триазола из азида и алкинила) второго адаптерного олигонуклеотида (например, одноцепочечной ДНК), содержащего универсальную последовательность С, с 3' концами фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для создания библиотеки для секвенирования нового поколения; определение нуклеотидной последовательности фрагмента библиотеки, представляющей собой «лестницу» фрагментов, (например, с использованием олигонуклеотидного праймера, комплементарного универсальной последовательности С), где указанная нуклеотидная последовательность содержит нуклеотидную субпоследовательность нуклеотидной последовательности-мишени; определение нуклеотидной последовательности-штрихкода фрагмента библиотеки, представляющей собой «лестницу» фрагментов (например, с использованием олигонуклеотидного праймера, комплементарного универсальной последовательности В); объединение нуклеотидной последовательности-штрихкода с источником нуклеотидной последовательности-мишени; группировку нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, имеющих одинаковую нуклеотидную последовательность-штрихкод; сборку множества нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для получения консенсусной последовательности и картирование консенсусной последовательность к референсной последовательности, где нуклеотидные последовательности фрагментов библиотеки, представляющей собой «лестницу» фрагментов, содержат от 15 до 50, от 15 до 40 или от 15 до 30 нуклеотидов, и соответствуют перекрывающимся нуклеотидным субпоследовательностям в пределах нуклеотидной последовательности-мишени, и 3' концы указанных нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, соответствуют разным нуклеотидам нуклеотидной последовательности-мишени, и консенсусная последовательность сохраняет информацию фазирования и/или сцепления нуклеиновой кислоты-мишени.
Некоторые варианты реализации изобретения относятся к способам, композициям, наборам и системам для секвенирования нуклеиновой кислоты (например, с помощью NGS) путем создания библиотеки для секвенирования нового поколения с использованием модифицированных нуклеотидов, например, одного или более 3'-О-модифицированных нуклеотидов, таких как 3'-О-алкинил-модифицированные нуклеотиды. Согласно некоторым вариантам реализации изобретения, 3'-О-модифицированные нуклеотиды представляют собой 3'-О-пропаргил-нуклеотиды (например, 3'-О-пропаргил-dNTP, например, 3'-О-пропаргил-dATP, 3'-О-пропаргил-dCTP, 3'-О-пропаргил-dGTP, 3'-О-пропаргил-dTTP; см., например, заявки на патенты США №14/463,412 и 14/463,416; и международную патентную заявку PCT/US 2014/051726, каждая из которых полностью включена в настоящую заявку посредством ссылки во всех отношениях). Например, варианты реализации технологии относятся к созданию библиотеки секвенирования (например, для NGS), содержащей «лестницу» фрагментов нуклеиновой кислоты, полученную путем включения терминирующих рост цепь 3'-О-модифицированных нуклеотидов с помощью полимеразы во время синтеза нуклеиновой кислоты in vitro.
Конкретные варианты реализации относятся к созданию «лестницы» фрагментов нуклеиновой кислоты с использованием полимеразной реакции, включающей стандартные dNTP и 3'-О-пропаргил-dNTP в молярном отношении, составляющем от 1:500 до 500:1 (например, отношение стандартных dNTP к 3'-О-пропаргил-dNTP составляет 1:500, 1:450, 1:400, 1:350, 1:300, 1:250, 1:200, 1:150, 1:100, 1:90, 1:80, 1:70, 1:60, 1:50, 1:40, 1:30, 1:20, 1:10, 1:9, 1:8, 1:7, 1:6, 1:5, 1:4, 1:3, 1:2, 2:1, 3:1, 4:1, 5:1, 6:1, 7:1, 8:1, 9:1, 10:1, 20:1, 30:1, 40:1, 50:1, 60:1, 70:1, 80:1, 90:1, 100:1, 150:1, 200:1, 250:1, 300а, 350:1, 400:1, 450:1 или 500:1). Терминированные фрагменты нуклеиновой кислоты, полученные с помощью способов, описанных в настоящей заявке, содержат пропаргильную группу на 3' концах. Дополнительные варианты реализации изобретения относятся к присоединению адаптера к 3' концам фрагментов нуклеиновой кислоты с использованием химического конъюгирования. Например, согласно некоторым вариантам реализации изобретения, 5'-азидо-модифицированный олигонуклеотид (например, 5'-азидо-метил-модифицированный олигонуклеотид) конъюгируют с 3'-пропаргил-терминированными фрагментами нуклеиновой кислоты с помощью реакций «клик-химии» (например, в реакции, катализируемой медью (например, реагентом на основе меди (I)). Согласно некоторым вариантам реализации изобретения, сначала амплифицируется область-мишень (например, с помощью ПЦР) с получением ампликона мишени для секвенирования. Согласно некоторым вариантам реализации изобретения, амплификация области-мишени включает амплификацию области-мишени с использованием от 5 до 15 циклов (например, с помощью «малоцикловой» амплификации).
Согласно дополнительным вариантам реализации изобретения ампликон мишени содержит метку (например, содержит штрихкод последовательность), например, ампликон мишени представляет собой поддающийся идентификации ампликон. Согласно некоторым вариантам реализации изобретения, праймер, используемый в амплификации области-мишени, содержит метку (например, штрихкод-последовательность), которая затем включается в ампликон мишени (например, в реакции «копирования и мечения») с получением поддающегося идентификации ампликона. Согласно некоторым вариантам реализации изобретения, адаптер, содержащий метку (например, содержащий штрихкод-последовательность), лигируют с ампликоном мишени после амплификации (например, в ходе лигазной реакции) с получением поддающегося идентификации комплекса адаптер-ампликон. Согласно некоторым вариантам реализации изобретения, 3' область праймера, используемого для получения поддающегося идентификации ампликона в ходе реакции копирования и мечения содержит специфичную по отношению к мишени праймирующую последовательность, и 5' область указанного праймера содержит две различные универсальные последовательности (например, универсальную последовательность А и универсальную последовательность В), фланкирующие вырожденную последовательность. Согласно некоторым вариантам реализации изобретения, адаптер, лигированный с ампликоном с образованием поддающегося идентификации комплекса адаптер-ампликон, представляет собой двуцепочечный адаптер, например, имеющий одну цепь, содержащую вырожденную последовательность (например, содержащую от 8 до 12 оснований), фланкированную как с 5' конца, так и с 3' конца разными универсальными последовательностями (например, универсальной последовательностью А и универсальной последовательностью В), и вторую цепь, содержащую универсальную последовательность С (например, на 5' конце) и последовательность (например, на 3' конце), которая комплементарна универсальной последовательности В и которая имеет дополнительный Τ в 3'-концевом положении.
Кроме того, варианты реализации технологии обеспечивают создание «лестницы» фрагментов нуклеиновой кислоты на основе комплекса адаптер-ампликон, например, для получения библиотеки для секвенирования NGS. В частности, технология обеспечивает создание «лестницы» 3'-О-пропаргил-dN-терминированных нуклеиновых кислот для секвенирования нуклеиновых кислот (например, NGS), например, с использованием полимеразной реакции, включающей стандартных dNTP и 3'-О-пропаргил-dNTP в молярном отношении, составляющем от 1:500 до 500:1 (отношение стандартных dNTP к 3'-О-пропаргил-dNTP). Кроме того, согласно некоторым вариантам реализации изобретения, технология обеспечивает присоединение адаптера к 3' концам фрагментов нуклеиновой кислоты с использованием химической конъюгации. Например, согласно некоторым вариантам реализации изобретения, 5'-азидо-модифицированный олигонуклеотид (например, 5'-азидо-метил-модифицированный олигонуклеотид) конъюгируют с 3'-пропаргил-терминированными фрагментами нуклеиновой кислоты с помощью реакций «клик-химии» (например, в ходе реакции, катализируемой медью (например, реагентом на основе меди (I))).
Таким образом, согласно некоторым вариантам реализации изобретения, предложен способ создания библиотеки для секвенирования нового поколения, включающий амплификацию нуклеотидной последовательности-мишени с использованием праймера, содержащего специфичную по отношению к мишени последовательность, универсальную последовательность А, универсальную последовательность В и нуклеотидную последовательность-штрихкод (например, содержащую от 1 до 20 нуклеотидов), связанную с нуклеиновой кислотой-мишенью, для получения поддающегося идентификации ампликона; создание «лестницы» фрагментов нуклеиновой кислоты на основе поддающегося идентификации ампликона с использованием содержащего 3'-О-блокирующую группу нуклеотидного аналога (например, 3'-О-алкинил-нуклеотидного аналога, 3'-О-пропаргил-нуклеотидного аналога); и лигирование (например, с помощью реакций «клик-химии», например, с использованием каталитического реагента на основе меди, например, для образования триазола из азида и алкинила) второго адаптерного олигонуклеотида (например, одноцепочечной ДНК), содержащего универсальную последовательность С, с 3' концами фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для создания библиотеки для секвенирования нового поколения, где нуклеотидные последовательности фрагментов библиотеки, представляющей собой «лестницу» фрагментов, содержат от 15 до 100 нуклеотидов и соответствуют перекрывающимся нуклеотидным субпоследовательностям в пределах нуклеотидной последовательности-мишени, и 3' концы указанных нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, соответствуют разным нуклеотидам нуклеотидной последовательности-мишени.
Согласно некоторым вариантам реализации изобретения, предложен способ создания библиотеки для секвенирования нового поколения, включающий амплификацию нуклеотидной последовательности-мишени для получения ампликона; лигирование адаптера (например, адаптера, содержащего одну цепь, содержащего вырожденную последовательность (например, содержащего от 8 до 12 оснований), фланкированную как на 5' конце, так и на 3' конце двумя разными универсальными последовательностями (например, универсальной последовательностью А и универсальной последовательностью В,) и вторую цепь, содержащую универсальную последовательность С (например, на 5' конце) и последовательность (например, на 3' конце), которая комплементарна универсальной последовательности В и содержит дополнительный Τ в 3'-концевом положении), с ампликоном с получением комплекса адаптер-ампликон; создание «лестницы» фрагментов нуклеиновой кислоты на основе комплекса адаптер-ампликон с использованием содержащего 3'-О-блокирующую группу нуклеотидного аналога (например, 3'-О-алкинил-нуклеотидного аналога, 3'-О-пропаргил-нуклеотидного аналога); и лигирование (например, с помощью реакций «клик-химии», например, с использованием каталитического реагента на основе меди, например, для образования триазола из азида и алкинила) второго адаптерного олигонуклеотида (например, одноцепочечной ДНК), содержащего универсальную последовательность С, с 3' концами фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для создания библиотеки для секвенирования нового поколения, где нуклеотидные последовательности фрагментов библиотеки, представляющей собой «лестницу» фрагментов, содержат от 15 до 100 нуклеотидов и соответствуют перекрывающимся нуклеотидным субпоследовательностям в пределах нуклеотидной последовательности-мишени, и 3' концы нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов соответствуют разным нуклеотидам нуклеотидной последовательности-мишени.
Согласно некоторым вариантам реализации изобретения, предложен способ определения нуклеотидной последовательности-мишени, включающий амплификацию нуклеотидной последовательности-мишени с использованием праймера, содержащего специфичную по отношению к мишени последовательность, универсальную последовательность А, универсальную последовательность В и нуклеотидную последовательность-штрихкод (например, содержащую от 1 до 20 нуклеотиды), связанную с нуклеиновой кислотой-мишенью, для получения поддающегося идентификации ампликона; создание «лестницы» фрагментов нуклеиновой кислоты на основе поддающегося идентификации ампликона с использованием содержащего 3'-О-блокирующую группу нуклеотидного аналога (например, 3'-О-алкинил-нуклеотидного аналога, 3'-О-пропаргил-нуклеотидного аналога); и лигирование (например, с помощью реакций «клик-химии», например, с использованием каталитического реагента на основе меди, например, для образования триазола из азида и алкинила) второго адаптерного олигонуклеотида (например, одноцепочечной ДНК), содержащего универсальную последовательность С, с 3' концами фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для создания библиотеки для секвенирования нового поколения; определение нуклеотидной последовательности фрагмента библиотеки, представляющей собой «лестницу» фрагментов, (например, с использованием олигонуклеотидного праймера, комплементарного универсальной последовательности С), где указанная нуклеотидная последовательность содержит нуклеотидную субпоследовательность нуклеотидной последовательности-мишени; определение нуклеотидной последовательности-штрихкода фрагмента библиотеки, представляющей собой «лестницу» фрагментов; объединение нуклеотидной последовательности-штрихкода с источником нуклеотидной последовательности-мишени; группировку нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, имеющих одинаковую нуклеотидную последовательность-штрихкод; сборку множества нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для получения консенсусный последовательности; и, согласно некоторым вариантам реализации изобретения, картирование консенсусной последовательности к референсной последовательности, где нуклеотидные последовательности фрагментов библиотеки, представляющей собой «лестницу» фрагментов, содержат от 15 до 50, от 15 до 40 или от 15 до 30 нуклеотидов и соответствуют перекрывающимся нуклеотидным субпоследовательностям в пределах нуклеотидной последовательности-мишени, и 3' концы указанных нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, соответствуют разным нуклеотидам нуклеотидной последовательности-мишени, и консенсусная последовательность сохраняет информацию фазирования и/или сцепления нуклеиновой кислоты-мишени.
Согласно некоторым вариантам реализации изобретения, предложен способ определения нуклеотидной последовательности-мишени, включающий амплификацию нуклеотидной последовательности-мишени для получения ампликона; лигирование адаптера (например, адаптера, содержащего одну цепь, содержащего вырожденную последовательность (например, содержащую от 8 до 12 оснований), фланкированную на 5' конце и 3' конце двумя различными универсальными последовательностями (например, универсальной последовательностью А и универсальной последовательностью В), и вторую цепь, содержащую универсальную последовательность С (например, на 5' конце) и последовательность (например, на 3' конце), которая комплементарна универсальной последовательности В и которая содержит дополнительный Τ в 3'-концевом положении, с ампликоном для получения комплекса адаптер-ампликон; создание «лестницы» фрагментов нуклеиновой кислоты на основе комплекса адаптер-ампликон с использованием содержащего 3'-О-блокирующую группу нуклеотидного аналога (например, 3'-О-алкинил-нуклеотидного аналога, 3'-О-пропаргил-нуклеотидного аналога); и лигирование (например, с помощью реакций «клик-химии», например, с использованием каталитического реагента на основе меди, например, для образования триазола из азида и алкинила) второго адаптерного олигонуклеотида (например, одноцепочечной ДНК), содержащего универсальную последовательность С, с 3' концами фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для создания библиотеки для секвенирования нового поколения; определение нуклеотидной последовательности фрагмента библиотеки, представляющей собой «лестницу» фрагментов (например, с использованием олигонуклеотидного праймера, комплементарного универсальной последовательности С), где указанная нуклеотидная последовательность содержит нуклеотидную субпоследовательность нуклеотидной последовательности-мишени; определение нуклеотидной последовательности-штрихкода фрагмента библиотеки, представляющей собой «лестницу» фрагментов; объединение нуклеотидной последовательности-штрихкода с источником нуклеотидной последовательности-мишени; группировку нуклеотидной последовательности фрагментов библиотеки, представляющей собой «лестницу» фрагментов, содержащих одинаковую нуклеотидную последовательность-штрихкод; сборку множества нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для получения консенсусной последовательности; и, согласно некоторым вариантам реализации изобретения, картирование консенсусной последовательности к референсной последовательности, где нуклеотидные последовательности фрагментов библиотеки, представляющей собой «лестницу» фрагментов, включают от 15 до 50, от 15 до 40 или от 15 до 30 нуклеотидов и соответствуют перекрывающимся нуклеотидным субпоследовательностям в пределах нуклеотидной последовательности-мишени, и 3' концы указанных нуклеотидных последовательностей фрагментов библиотеки, представляющей собой «лестницу» фрагментов, соответствуют разным нуклеотидам нуклеотидной последовательности-мишени, и консенсусная последовательность сохраняет информацию фазирования и/или сцепления нуклеиновой кислоты-мишени.
Согласно некоторым вариантам реализации изобретения, предложен способ определения нуклеотидной последовательности-мишени, включающий определение первой нуклеотидной субпоследовательности нуклеотидной последовательности-мишени (например, путем праймирования универсальной последовательностью и, например, терминации полимеризации с помощью содержащего 3'-О-блокирующую группу нуклеотидного аналога, такого как 3'-О-алкинил-нуклеотидный аналог или 3'-О-пропаргил-нуклеотидный аналог, или терминации полимеризации с помощью нуклеотидного аналога, содержащего обратимый терминатор), где 5' конец указанной первой нуклеотидной субпоследовательности находится на нуклеотиде x1 нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде y1 нуклеотидной последовательности-мишени; определение второй нуклеотидной субпоследовательности нуклеотидной последовательности-мишени (например, путем праймирования универсальной последовательностью и, например, терминации полимеризации с помощью содержащего 3'-О-блокирующую группу нуклеотидного аналога, такого как 3'-О-алкинил-нуклеотидный аналог или 3'-О-пропаргил-нуклеотидный аналог или терминации полимеризации с помощью нуклеотидного аналога, содержащего обратимый терминатор), где 5' конец указанной второй нуклеотидной субпоследовательности находится на нуклеотиде х2 нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде у2 нуклеотидной последовательности-мишени; сборку первой нуклеотидной субпоследовательности и второй нуклеотидной субпоследовательности для получения консенсусной последовательности (например, содержащей 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000 или более чем 1000, например, 2000, 2500, 3000, 3500, 4000, 4500 или 5000 или более чем 5000 оснований) для нуклеотидной последовательности-мишени; идентификацию источника или образца нуклеотидной последовательности-мишени путем декодирования нуклеотидной последовательности-штрихкода; картирование консенсусной последовательности (например, сохраняющей информацию фазирования и/или сцепления нуклеиновой кислоты-мишени) к референсной последовательности, где х2<y1; и (y1-x1)<100 (например, (y1-x1)<90, 80, 70, 60, 55, 50, 45, 40, 35 или 30), (у2-х2)<100 (например, (y1-x1)<90, 80, 70, 60, 55, 50, 45, 40, 35 или 30), и (у2-у1)<20 (например, (у2-у1)<10, (у2-у1)<5, (у2-у1)<4, (у2-y1)<3, (у2-y1)<2, или (у2-y1=1).
Согласно некоторым вариантам реализации изобретения, предложен способ определения нуклеотидной последовательности-мишени, включающий определение n нуклеотидных субпоследовательностей нуклеотидной последовательности-мишени (например, путем праймирования универсальной последовательностью и, например, терминации полимеризации с помощью содержащего 3'-О-блокирующую группу нуклеотидного аналога, такого как 3'-О-алкинил-нуклеотидный аналог или 3'-О-пропаргил-нуклеотидный аналог, или терминации полимеризации с помощью нуклеотидного аналога, содержащего обратимый терминатор), где 5' конец нуклеотидной субпоследовательности m находится на нуклеотиде xm нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде ym нуклеотидной последовательности-мишени; и 5' конец нуклеотидной субпоследовательности (m+1) находится на нуклеотиде xm+1 нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде ym+1 нуклеотидной последовательности-мишени; сборку n нуклеотидных субпоследовательностей для получения консенсусной последовательности (например, содержащей 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000 или более чем 1000 оснований, например, 2000, 2500, 3000, 3500, 4000, 4500 или 5000 или более чем 5000 оснований) для нуклеотидной последовательности-мишени; идентификацию источника или образца нуклеотидной последовательности-мишени путем декодирования нуклеотидной последовательности-штрихкод; и картирование консенсусной последовательности к референсной последовательности, где: m варьирует от 1 до n, xm+1<ym; и (ym-xm)<100 (например, (ym-xm)<90, 80, 70, 60, 55, 50, 45, 40, 35 или 30), (ym+1-xm+1)<100 (например, (ym+1-xm+1)<90, 80, 70, 60, 55, 50, 45, 40, 35 или 30), и (ym+1-ym)<20 (например, (ym+1-ym)<10, (ym+1-ym)<5, (ym+1-ym)<4, (ym+1-ym)<3 или (ym+1-ym)=1), и консенсусная последовательность сохраняет информацию фазирования и/или сцепления нуклеиновой кислоты-мишени.
Некоторые варианты реализации технологии обеспечивают композицию для применения в качестве библиотеки для секвенирования нового поколения для получения последовательности нуклеиновой кислоты-мишени, содержащую 3'-О-заблокированный нуклеотидный аналог, 3'-О-алкинил-нуклеотидный аналог, 3'-О-пропаргил-нуклеотидный аналог или нуклеотидный аналог, содержащий обратимый терминатор; праймер для секвенирования (например, комплементарный универсальной последовательности С); второй праймер для секвенирования (например, комплементарный универсальной последовательности В); и n нуклеиновых кислот, содержащих 3'-О-заблокированный нуклеотидный аналог, 3'-О-алкинил-нуклеотидный аналог или 3'-О-пропаргил-нуклеотидный аналог, связанный (например, триазольной связью, образованной, например, с помощью реакций «клик-химии», например, с помощью реакции между азидом и алкилом, катализируемой катализатором на основе меди) с адаптером (например, адаптерным олигонуклеотидом для секвенирования нового поколения) или нуклеотидным аналогом, содержащим обратимый терминатор, где каждая нуклеиновая кислота содержит нуклеотидную субпоследовательность нуклеиновой кислоты-мишени, универсальную последовательность В, содержащую от 10 до 100 нуклеотидов, универсальную последовательность С, содержащую от 10 до 100 нуклеотидов и/или нуклеотидную последовательность-штрихкод, содержащую от 1 до 20 нуклеотидов, где 5' конец нуклеотидной субпоследовательности m находится на нуклеотиде xm нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде ym нуклеотидной последовательности-мишени; 5' конец нуклеотидной субпоследовательности (m+1) находится на нуклеотиде xm+1 нуклеотидной последовательности-мишени, и 3' конец указанной нуклеотидной субпоследовательности находится на нуклеотиде ym+1 нуклеотидной последовательности-мишени; m варьирует от 1 до n; xm=xm+1; (ym+1-ym)<20 (например, (ym+1-ym)<15, (ym+1-ym)<10, (ym+1-ym)<5, (ym+1-ym)<4, (ym+1-ym)<3 или (ym+1-ym)=1); n нуклеиновые кислоты содержат нуклеиновые кислоты, содержащие разные нуклеотидные последовательности-штрихкоды и разные нуклеотидные субпоследовательности нуклеотидной последовательности-мишени, где каждая нуклеотидная последовательность-штрихкод связана (например, с абсолютным соответствием) с нуклеотидной последовательностью-мишенью.
Некоторые варианты реализации технологии обеспечивают композицию для применения в качестве библиотеки для секвенирования нового поколения для получения последовательности нуклеиновой кислоты-мишени, содержащую n нуклеиновых кислот (например, библиотеку фрагментов нуклеиновых кислот), где каждая из n нуклеиновых кислот содержит содержащий 3'-О-блокирующую группу нуклеотидный аналог (например, 3'-О-алкинил-нуклеотидный аналог, такой как 3'-О-пропаргил-нуклеотидный аналог). Согласно некоторым вариантам реализации изобретения, каждая нуклеиновая кислота из n нуклеиновых кислот содержит нуклеотидную субпоследовательность нуклеотидной последовательности-мишени. В частности, варианты реализации изобретения обеспечивают композицию, содержащую n нуклеиновых кислот, где каждая из n нуклеиновых кислот терминирована с помощью содержащего 3'-О-блокирующую группу нуклеотидного аналога (например, 3'-О-алкинил-нуклеотидного аналога, такого как 3'-О-пропаргил-нуклеотидный аналог). Дополнительные варианты реализации изобретения обеспечивают композицию, содержащую n нуклеиновых кислот (например, библиотеку фрагментов нуклеиновых кислот), где каждая из n нуклеиновых кислот содержит 3'-О-заблокированный нуклеотидный аналог (например, 3'-О-алкинил-нуклеотидный аналог, такой как 3'-О-пропаргил-нуклеотидный аналог), и каждая из n нуклеиновых кислот конъюгирована (например, связана) с олигонуклеотидным адаптером с помощью триазольной связи (например, связи, образованной в результате химического конъюгирования пропаргильной группы и азидо группы, например, с помощью реакции «клик-химии»). Например, согласно некоторым вариантам реализации изобретения, предложена композиция, содержащая n нуклеиновых кислот (например, библиотеку фрагментов нуклеиновых кислот), где каждая из n нуклеиновых кислот содержит 3'-О-пропаргил-нуклеотидный аналог (например, 3'-О-пропаргил-dA, 3'-О-пропаргил-dC, 3'-О-пропаргил-dG, и/или 3'-О-пропаргил-dT), конъюгированный (например, связанный) с олигонуклеотидным адаптером с помощью триазольной связи (например, связи, образованной в результате химического конъюгирования пропаргильной группы и азидо группы, например, с помощью реакции «клик-химии»).
Согласно некоторым вариантам реализации изобретения, композицию для применения в качестве библиотеки для секвенирования нового поколения для получения последовательности нуклеиновой кислоты-мишени получают с помощью способа, включающего синтез n нуклеиновых кислот (например, библиотеки фрагментов нуклеиновых кислот) с использованием смеси dNTP и одного или более содержащих 3'-О-блокирующую группу нуклеотидных аналогов (аналога) (например, одного или более 3'-О-алкинил-нуклеотидных аналогов (аналога), таких как один или более 3'-О-пропаргил-нуклеотидных аналогов (аналога)), например, в молярном отношении, составляющем от 1:500 до 500:1 (например, 1:500, 1:450, 1:400, 1:350, 1:300, 1:250, 1:200, 1:150, 1:100, 1:90, 1:80, 1:70, 1:60, 1:50, 1:40, 1:30, 1:20, 1:10, 1:9, 1:8, 1:7, 1:6, 1:5, 1:4, 1:3, 1:2, 2:1, 3:1, 4:1, 5:1, 6:1, 7:1, 8:1, 9:1, 10:1, 20:1, 30:1, 40:1, 50:1, 60:1, 70:1, 80:1, 90:1, 100:1, 150:1, 200:1, 250:1, 300:1, 350:1, 400:1, 450:1 или 500:1). Согласно некоторым вариантам реализации изобретения, композицию получают с использованием полимеразы, полученной, произошедшей, выделенной, клонированной и т.д. из видов рода Thermococcus (например, организма таксономической группы Archaea; Euryarchaeota; Thermococci; Thermococcales; Thermococcaceae; Thermococcus). Согласно некоторым вариантам реализации изобретения, полимераза получена, происходит, выделена, клонирова и т.д. из вида Thermococcus 9°N-7. Согласно некоторым вариантам реализации изобретения, полимераза содержит аминокислотные замены, которые обеспечивают улучшенное включение модифицированных субстратов, таких как модифицированные дидезоксинуклеотиды, рибонуклеотиды и ациклонуклеотиды. Согласно некоторым вариантам реализации изобретения, полимераза содержит аминокислотные замены, которые обеспечивают улучшенное включение нуклеотидных аналогов, содержащих модифицированные 3' функциональные группы, таких как 3'-О-пропаргил dNTP, описанные в настоящей заявке. Согласно некоторым вариантам реализации изобретения, аминокислотная последовательность полимеразы содержит одну или более аминокислотных замен по сравнению с аминоксилотной последовательностью полимеразы Thermococcus sp. 9°N-7 дикого типа, например, замену аспарагиновой кислоты на аланин в положении аминокислоты 141 (D141A), замену глутаминовой кислоты на аланин в положении аминокислоты 143 (Е143А), замену тирозина на валин в положении аминокислоты 409 (Y409V) и/или замену аланина на лейцин в положении аминокислоты 485 (A485L). Согласно некоторым вариантам реализации изобретения, полимераза обеспечивается в гетерологичном организме-хозяине, таком как Escherichia coli, который содержит клонированный ген полимеразы Thermococcus sp. 9°N-7, например, содержащий одну или более мутаций (например, D141A, Е143А, Y409V и/или A485L). Согласно некоторым вариантам реализации изобретения, полимераза представляет собой полиемразу Thermococcus sp. 9°N-7, продаваемую под торговой маркой THERMINATOR (например, THERMINATOR II) компанией New England BioLabs (Ипсвич, Массачусетс).
Таким образом, технология относится к реакционным смесям, содержащим нуклеиновую кислоту-мишень, смеси dNTP и один или более содержащих 3'-О-блокирующую группу нуклеотидных аналогов (аналога) (например, один или более 3'-О-алкинил-нуклеотидных аналогов (аналога), таких как один или более 3'-О-пропаргил-нуклеотидных аналогов (аналога)), например, в молярном отношении, составляющем от 1:500 до 500:1 (например, 1:500, 1:450, 1:400, 1:350, 1:300, 1:250, 1:200, 1:150, 1:100, 1:90, 1:80, 1:70, 1:60, 1:50, 1:40, 1:30, 1:20, 1:10, 1:9, 1:8, 1:7, 1:6, 1:5, 1:4, 1:3, 1:2, 2:1, 3:1, 4:1, 5:1, 6:1, 7:1, 8:1, 9:1, 10:1, 20:1, 30:1, 40:1, 50:1, 60:1, 70:1, 80:1, 90:1, 100:1, 150:1, 200:1, 250:1, 300:1, 350:1, 400:1, 450:1 или 500:1), и полимеразу для синтеза нуклеиновой кислоты с использованием dNTP и одного или более 3'-О-заблокированных нуклеотидных аналогов (аналога) (например, полимеразу, полученную, произошедшую, выделенную, клонированную и т.д. из видов рода Thermococcus). Согласно некоторым вариантам реализации изобретения, нуклеиновая кислота-мишень представляет собой ампликон. Согласно некоторым вариантам реализации изобретения, нуклеиновая кислота-мишень содержит штрихкод. Согласно некоторым вариантам реализации изобретения, нуклеиновая кислота-мишень представляет собой ампликон, содержащий штрихкод. Согласно некоторым вариантам реализации изобретения, нуклеиновая кислота-мишень представляет собой ампликон, лигированный с адаптером, содержащим штрихкод. Согласно некоторым вариантам реализации изобретения, предложены реакционные смеси, которые содержат множество нуклеиновых кислот-мишеней, где каждая нуклеиновая кислота-мишень, содержит штрихкод, связанный с идентифицирующим признаком указанной нуклеиновой кислоты-мишени.
Согласно некоторым вариантам реализации изобретения, предложена композиция реакционной смеси, содержащая матрицу (например, кольцевую матрицу, например, содержащую универсальную нуклеотидной последовательность и/или нуклеотидную последовательность-штрихкод), содержащая субпоследовательность нуклеиновой кислоты-мишени, полимеразу, один или более фрагментов библиотеки, представляющей собой «лестницу» фрагментов, и 3'-О-заблокированный нуклеотидный аналог.
Согласно некоторым вариантам реализации изобретения, предложена композиция реакционной смеси, содержащая библиотеку нуклеиновых кислот, где указанная библиотека нуклеиновых кислот содержит перекрывающиеся короткие нуклеотидные последовательности, покрывающие нуклеиновую кислоту-мишень (например, перекрывающиеся короткие нуклеотидные последовательности покрывают область нуклеиновой кислоты-мишени, содержащую 100 оснований, 200 оснований, 300 оснований, 400 оснований, 500 оснований, 600 оснований, 700 оснований, 800 оснований, 900 оснований, 1000 оснований или более чем 1000 оснований, например, 2000 оснований, 2500 оснований, 3000 оснований, 3500 оснований, 4000 оснований, 4500 оснований, 5000 оснований или более чем 5000 оснований) и смещенные друг относительно друга на 1-20, 1-10 или 1-5 оснований (например, на 1 основание), и каждая нуклеиновая кислота библиотеки содержит менее чем 100 оснований, менее чем 90 оснований, менее чем 80 оснований, менее чем 70 оснований, менее чем 60 оснований, менее чем 50 оснований, менее чем 45 оснований, менее чем 40 оснований, менее чем 35 оснований или менее чем 30 оснований.
Согласно некоторым вариантам реализации изобретения, предложен набор для создания библиотеки для секвенирования, где указанный набор содержит адаптерный олигонуклеотид, содержащий первую реакционноспособную группу (например, азид), 3'-О-заблокированный нуклеотидный аналог (например, 3'-О-алкинил-нуклеотидный аналог или 3'-О-пропаргил-нуклеотидный аналог, например, содержащий алкиновую группу, например, содержащий вторую реакционноспособную группу, которая образует химическую связь с первой реакционноспособной группой, например, с использованием реакций «клик-химии»), полимеразу (например, полимеразу для изотермической амплификации или термоциклирования), второй адаптерный олигонуклеотид, одну или более композиций, содержащих нуклеотид или смесь нуклеотидов, и лигазу или каталитический реагент на основе меди для реакций «клик-химии».
Согласно некоторым вариантам реализации наборов, указанные наборы содержат один или более 3'-О-заблокированных нуклеотидных аналогов (аналог) (например, однин или более 3'-О-алкинил-нуклеотидных аналогов (аналог), таких как один или более 3'-О-пропаргил-нуклеотидных аналогов (аналог) и один или более адаптерных олигонуклеотидов, содержащих азидную группу (например, 5'-азидо олигонуклеотид, например, 5'-азидо-метил-олигонуклеотид). Некоторые варианты реализации наборов также обеспечивают 5'-азидо-метил-олигонуклеотид, содержащий штрихкод. Некоторые варианты реализации наборов дополнительно обеспечивают множество 5'-азидо-метилолигонуклеотидов, содержащих множество штрихкодов (например, каждый 5'-азидо-метил-олигонуклеотид содержит штрихкод, который отличается от одного или более других штрихкодов одного или более других 5'-азидо-метил олигонуклеотидов (олигонуклеотида), содержащих другой штрихкод). Дополнительные варианты реализации наборов включают каталитический реагент для реакций «клик-химии» (например, каталитический реагент на основе меди (I)).
Некоторые варианты реализации наборов включают один или более стандартных dNTP помимо одного или более 3'-О-заблокированных нуклеотидных аналогов (аналога) (например, одного или более 3'-О-алкинил-нуклеотидных аналогов (аналога), например, одного или более 3'-О-пропаргил-нуклеотидных аналогов (аналога)). Например, некоторые варианты реализации наборов обеспечивают dATP, dCTP, dGTP и dTTP в отдельной емкости или в виде смеси с одним или более 3'-О-пропаргил-dATP, 3'-О-пропаргил-dCTP, 3'-О-пропаргил-dGTP и/или 3'-О-пропаргил-dATP.
Некоторые варианты реализации наборов дополнительно содержат полимеразу, полученную, произошедшую, выделенную, клонированную и т.д. из видов рода Thermococcus (например, из организма таксономической группы Archaea; Euryarchaeota; Thermococci; Thermococcales; Thermococcaceae; Thermococcus). Согласно некоторым вариантам реализации изобретения, полимераза получена, произошла, выделена, клонирова и т.д. из вида Thermococcus 9°N-7. Согласно некоторым вариантам реализации изобретения, полимераза содержит аминокислотные замены, которые обеспечивают улучшенное включение модифицированных субстратов, таких как модифицированные дидезоксинуклеотиды, рибонуклеотиды и ациклонуклеотиды. Согласно некоторым вариантам реализации изобретения, полимераза содержит аминокислотные замены, которые обеспечивают улучшенное включение нуклеотидных аналогов, содержащих модифицированные 3' функциональные группы, таких как 3'-О-пропаргил dNTP, описанные в настоящей заявке. Согласно некоторым вариантам реализации изобретения, аминокислотная последовательность полимеразы содержит одну или более аминокислотных замен по сравнению с аминоксилотной последовательностью полимеразы дикого типа Thermococcus sp. 9°N-7, например, замену аспарагиновой кислоты на аланин в положении аминокислоты 141 (D141A), замену глутаминовой кислоты на аланин в положении аминокислоты 143 (Е143А), замену тирозина на валин в положении аминокислоты 409 (Y409V) и/или замену лейцина на аланин в положении аминокислоты 485 (A485L). Согласно некоторым вариантам реализации изобретения, полимераза обеспечивается в гетерологичном организме-хозяине, таком как Escherichia coli, который содержит клонированный ген полимеразы Thermococcus sp. 9°N-7, например, содержащий одну или более мутаций (например, D141A, Е143А, Y409V и/или A485L). Согласно некоторым вариантам реализации изобретения, полимераза представляет собой полимеразу Thermococcus sp. 9°N-7, продаваемую под торговым названием THERMINATOR (например, THERMINATOR II) компанией New England BioLabs (Ипсвич, Массачусетс).
Таким образом, некоторые варианты реализации наборов включают один или более 3'-О-пропаргил-нуклеотидных аналогов (аналога) (например, один или более 3'-О-пропаргил-dATP, 3'-О-пропаргил-dCTP, 3'-О-пропаргил-dGTP и/или 3'-О-пропаргил-dATP), смесь стандартных dNTP (например, dATP, dCTP, dGTP и dTTP), один или более 5'-азидо-метил-олигонуклеотидных адаптеров, полимеразу, полученную, произошедшую, выделенную, клонированную и т.д. из видов рода Thermococcus, и катализатор реакций «клик-химии» для образования триазола из азидной группы и алкильной группы. Согласно некоторым вариантам реализации изобретения, один или более 3'-О-пропаргил-нуклеотидных аналогов (аналога) (например, один или более 3'-О-пропаргил-dATP, 3'-О-пропаргил-dCTP, 3'-О-пропаргил-dGTP и/или 3'-О-пропаргил-dATP) и смесь стандартных dNTP (например, dATP, dCTP, dGTP и dTTP) представлены вместе, например, набор содержит раствор, содержащий один или более 3'-О-пропаргил-нуклеотидных аналогов (аналога) (например, один или более 3'-О-пропаргил-dATP, 3'-О-пропаргил-dCTP, 3'-О-пропаргил-dGTP и/или 3'-О-пропаргил-dATP) и смесь стандартных dNTP (например, dATP, dCTP, dGTP и dTTP). Согласно некоторым вариантам реализации изобретения, раствор содержит один или более 3'-О-пропаргил-нуклеотидных аналогов (аналога) (например, один или более 3'-О-пропаргил-dATP, 3'-О-пропаргил-dCTP, 3'-О-пропаргил-dGTP и/или 3'-О-пропаргил-dATP) и смесь стандартных dNTP (например, dATP, dCTP, dGTP и dTTP) в отношении, составляющем от 1:500 до 500:1 (например, 1:500, 1:450, 1:400, 1:350, 1:300, 1:250, 1:200, 1:150, 1:100, 1:90, 1:80, 1:70, 1:60, 1:50, 1:40, 1:30, 1:20, 1:10, 1:9, 1:8, 1:7, 1:6, 1:5, 1:4, 1:3, 1:2, 2:1, 3:1, 4:1, 5:1, 6:1, 7:1, 8:1, 9:1, 10:1, 20:1, 30:1, 40:1, 50:1, 60:1, 70:1, 80:1, 90:1, 100:1, 150:1, 200:1, 250:1, 300:1, 350:1, 400:1, 450:1 или 500:1).
Некоторые варианты реализации наборов дополнительно включают программу для обработки данных последовательности, например, для извлечения данных нуклеотидной последовательности из данных, полученных секвенатором; для идентификации штрихкодов и субпоследовательностей-мишеней на основе данных, полученных секвенатором; для выравнивания и/или сборки субпоследовательностей на основе данных, полученных секвенатором, для получения консенсусной последовательности и/или для выравнивания субпоследовательностей и/или консенсусной последовательности к референсной последовательности (например, для идентификации различий в последовательностях (например, для идентификации аллелей, гомологов, филогенетических отношений, хромосом, подобий или различий по последовательностям, мутаций и/или ошибок секвенирования и т.д.) и/или для исправления искажений последовательности (например, ошибок секвенирования).
Согласно некоторым вариантам реализации изобретения, предложена система для секвенирования нуклеиновой кислоты-мишени, где указанная система включает адаптерный олигонуклеотид, содержащий первую реакционноспособную группу (например, азид), 3'-О-заблокированный нуклеотидный аналог (например, 3'-О-алкинил-нуклеотидный аналог или 3'-О-пропаргил-нуклеотидный аналог, например, содержащий алкиновую группу и, например, содержащий вторую реакционноспособную группу, которая образует химическую связь с первой реакционноспособной группой, например, с использованием реакций «клик-химии», например, с использованием катализатора для реакций «клик-химии» на основе меди), устройство для секвенирования, «лестницу» фрагментов нуклеиновой кислоты (например, содержащую множество нуклеиновых кислот, 3' концы которых различаются менее чем на 20 нуклеотидов, менее чем на 10 нуклеотидов, менее чем на 5 нуклеотидов, менее чем на 4 нуклеотида, менее чем 3 нуклеотида или на 1 нуклеотид), и программу для сборки коротких перекрывающихся нуклеотидных последовательностей в консенсусную последовательность, где каждая короткая нуклеотидная последовательность содержит менее чем 100, менее чем 90, менее чем 80, менее чем 70, менее чем 60, менее чем 50, менее чем 45, менее чем 40, менее чем 35 или менее чем 30 оснований; короткие нуклеотидные последовательности, покрывающие нуклеиновую кислоту-мишень, содержат по меньшей мере 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 2000, 2500, 3000, 3500, 4000, 5000 или более чем 5000 оснований; и указанные короткие нуклеотидные последовательности смещены друг относительно друга на 1-20, 1-10 или 1-5 оснований.
Дополнительные варианты реализации изобретения будут очевидны специалистам в соответствующей области техники на основе знаний, предложенных в настоящей заявке.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
Указанные и другие признаки, аспекты и преимущества технологии согласно настоящему изобретению лучше понимаются со ссылкой на следующие фигуры:
Фигура 1 представляет собой схематичное изображение варианта реализации технологии для секвенирования нуклеиновой кислоты.
Фигура 2 представляет собой схематичное изображение варианта реализации технологии для получения библиотеки для секвенирования нового поколения. На Фигуре 2А показан один вариант реализации технологии, и на Фигуре 2В показан другой вариант реализации технологии. На Фигуре 2С показан другой вариант реализации технологии.
Фигура 3 представляет собой схематичное изображение варианта реализации технологии для секвенирования нуклеиновой кислоты.
Фигура 4 представляет собой схематичное изображение варианта реализации технологии для секвенирования нуклеиновой кислоты.
На Фигуре 5 показаны схемы процессов, относящихся к вариантам реализации технологии, которые могут применяться в секвенировании нуклеиновой кислоты. Фигура 5А представляет собой схему процесса, описывающего вариант реализации технологии, включающий получение данных последовательности из библиотеки для NGS и выявление перекрывающихся субпоследовательностей последовательности-мишени. Фигура 5В представляет собой схему процесса, описывающего вариант реализации технологии для извлечения объединяющих данные последовательностей файлов, содержащих данные последовательности, идентификации и выявления последовательности-мишени и выравнивания последовательностей-мишеней для получения консенсусной последовательности.
На Фигуре 6 показано предсказанное и экспериментальное покрытие последовательности-мишени короткими прочтениями последовательности, полученными с помощью вариантов реализации технологии. На Фигуре 6А показано выравнивание последовательности, состоящей из прочтений из 40 п.о., соответствующее профилю покрытия последовательности. Также показаны консенсусные и референсные последовательности (последовательность, состоящая из 177 п.о., содержащая экзон 2 человеческого гена RAS и частичного фланкирующая интронные последовательности). На Фигуре 6В показано предсказанное выравнивание коротких прочтений последовательности и соответствующий профиль покрытия последовательности для теоретической матричной референсной последовательности.
На Фигуре 7 показана схема варианта реализации технологии, связанного со схемой «копирования и мечения» с использованием полимеразного удлинения праймера, содержащего штрихкод-последовательность и универсальные последовательности.
На Фигуре 8 показана схема экспериментальной делеции продуктов реакции «копирования и мечения» и оценки эффективности блокатора полимеразного удлинения.
На Фигуре 9 показана схема способа, основанного на лигировании с адаптером молекулярного штрихкодирования, согласно конкретным вариантам реализации технологии.
На Фигуре 10 показана схема экспериментального выявления продуктов лигирования с адаптером.
На Фигуре 11 показана схема внутримолекулярного лигирования (циркуляризации) одноцепочечной ДНК как этапа в создании «лестницы» фрагментов согласно технологии, предложенной в настоящей заявке.
На Фигуре 12 показана схема экспериментального выявления кольцевых матриц, связанного с вариантами реализации технологии, относящимися к получению кольцевых матриц для получения «лестницы» фрагментов.
Необходимо понимать, что фигуры не обязательно изображены в масштабе, и объекты на фигурах не обязательно изображены в масштабе по отношению друг к другу. Фигуры представляют собой изображения, которые, как предполагается, вносят ясность и облегчают понимание различных вариантов реализации устройств, систем и способов, описанных в настоящей заявке. По возможности используются одинаковые референсные количества для всех изображений для ссылки на одинаковые или подобные части. Более того, следует понимать, что изображения никоим образом не ограничивают объем идей настоящего изобертения.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Технология в целом относится к получению нуклеотидной последовательности, такой как консенсусная последовательность или последовательность гаплотипа. Согласно некоторым вариантам реализации изобретения, в настоящей заявке предложена технология получения библиотеки коротких перекрывающихся фрагментов ДНК из более большого фрагмента ДНК-мишени, подлежащего секвенированию. Длины коротких перекрывающихся фрагментов ДНК находятся в таком диапазоне, что один фрагмент отличается от другого фрагмента на 1-5 оснований, предпочтительно на 1 основание, на 3' конце (например, фрагменты «лестницы», подобные фрагментам, полученным с помощью стандартных способов секвенирования по Сэнджеру). Согласно некоторым вариантам реализации изобретения, короткие перекрывающиеся фрагменты ДНК индексируют для создания библиотеки для секвенирования нового поколения (NGS). Библиотека применяется для проведения NGS путем инициации реакции секвенирования от варьирующих 3' концов фрагментов ДНК. Получение прочтений последовательности, содержащих от ~30 оснований до ~50 оснований, с 3' концов коротких перекрывающихся фрагментов, приводит к получению группы следующих друг за другом прочтений последовательности, содержащих от ~30 до ~50 оснований, покрывающих более длинную ДНК-мишень, подлежащую секвенированию, и смещенных друг относительно друга на 1-5 оснований, предпочтительно на 1 основание. Сборка перекрывающихся коротких прочтений последовательности, состоящих из ~30-50 п.о., приводит к получению длинного непрерывного прочтения, покрывающего более длинный участок (~800-1000 п.о.) фрагмента ДНК-мишени. Таким образом, каждое прочтение последовательности является результатом определения оснований наивысшего качества с помощью NGS (например, первых 20-100 оснований), и каждая последовательность оснований является консенсусной и состоит из 30-50 независимых прочтений последовательности высокого качества.
В описании указанной технологии заголовки разделов, используемые в настоящей заявке, приведены только в целях организации информации и никоим образом не ограничивают описанный объект изобретения.
В настоящем подробном описании различных вариантов реализации в целях объяснения перечислено множество конкретных деталей для обеспечения ясного понимания описанных вариантов реализации. Специалисту в данной области техники очевидно, однако, что указанные различные варианты реализации можно осуществлять на практике с указанными конкретными деталями или без них. В других примерах структуры и устройства показаны в виде блок-схем. Кроме того, специалист в данной области техники легко определит, что конкретный порядок описания и осуществления способов является иллюстративными и может варьировать в пределах сущности и объема различных вариантов реализации, описанных в настоящей заявке.
Все литературные и подобные материалы, цитированные в настоящей заявке, включая, но не ограничиваясь указанными, патенты, патентные заявки, статьи, книги, монографии и интернет-страницы, конкретно и полностью включены посредством ссылки во всех отношениях. Если иное не указано, все технические и научные термины, используемые в настоящей заявке, имеют значение, общепринятое специалистами в области техники, к которой относятся различные варианты реализации, описанные в настоящей заявке. Когда определения терминов во включенных ссылках отличаются от определений, предложенных согласно настоящему изобретению, определения, предложенные согласно настоящему изобретению, имеют преимущество.
Определения
Для лучшего понимания технологии согласно настоящему изобретению ниже приведены определения ряда терминов и фраз. Дополнительные определения представлены в подробном описании изобретения.
В настоящей заявке и формуле изобретения следующие термины имеют значения, однозначно определенные в настоящей заявке, если иное явно не следует из контекста. Фраза «согласно одному варианту реализации изобретения» при использовании в настоящей заявке может относиться к одному и тому же варианту реализации изобретения, но не обязательно относится к одному и тому же варианту реализации изобретения. Более того, фраза «согласно другому варианту реализации изобретения» при использовании в настоящей заявке может относиться, но не обязательно относится к разным вариантам реализации. Таким образом, как описано ниже, различные варианты реализации изобретения можно легко комбинировать в пределах сущности и объема изобретения.
Кроме того, при использовании в настоящей заявке термин «или» представляет собой инклюзивный оператор «или» и является эквивалентным термину «и/или», если иное явно не следует из контекста. Термин «основан на» не является эксклюзивным и позволяет быть основанным на неописанных дополнительных факторах, если иное явно не следует из контекста. Кроме того, в настоящей заявке ссылка на предмет в единственном числе включает указанный предмет во множественном числе. Предлог «в» включает значение «в» и «на».
При использовании в настоящей заявке, «нуклеотид» содержит «основание» (альтернативно, «нуклеиновое основание» или «нитрогенное основание»), «сахар» (в частности, сахар, содержащий пять атомов углерода, например, рибозу или 2-дезоксирибозу) и «фосфатный фрагмент» одной или более фосфатных групп (например, монофосфат, дифосфат или трифосфат, состоящий из одного, двух или трех связанных фосфатных групп соответственно). Без фосфатного фрагмента нуклеиновое основание и сахар представляют собой «нуклеозид». Нуклеотид, таким образом, также может называться нуклеозидмонофосфатом или нуклеозиддифосфатом или нуклеозидтрифосфатом в зависимости от числа присоединенных фосфатных групп. Фосфатный фрагмент, как правило, присоединен к 5-атому углерода сахара, несмотря на то что некоторые нуклеотиды включают фосфатные фрагменты, присоединенные к 2-атому углерода или 3-атому углерода сахара. Нуклеотиды содержат пурин (в нуклеотидах аденине и гуанине) или пиримидиновое основание (в нуклеотидах цитозине, тимине и урациле). Рибонуклеотиды представляют собой нуклеотиды, в которых сахар представляет собой рибозу. Дезоксирибонуклеотиды представляют собой нуклеотиды, в которых сахар представляет собой дезоксирибозу.
При использовании в настоящей заявке, термин «нуклеиновая кислота» означает любую молекулу нуклеиновой кислоты, включая, но не ограничиваясь указанными, ДНК, РНК и их гибриды. Основания нуклеиновой кислоты, которые образуют молекулы нуклеиновой кислоты, могут представлять собой основания А, С, G, Τ и U, а также их производные. Производные указанных оснований хорошо известны в данной области техники. Необходимо понимать, что термин включает эквиваленты и аналоги ДНК или РНК, полученные из нуклеотидных аналогов. При использовании в настоящей заявке указанный термин также включает кДНК, которая комплементарна или является копией ДНК, полученной на основе РНК-матрицы, например, в результате действия обратной транскриптазы.
При использовании в настоящей заявке, «данные секвенирования нуклеиновой кислоты», «информация секвенирования нуклеиновой кислоты», «последовательность нуклеиновой кислоты», «геномная последовательность», «генетическая последовательность», «фрагмент последовательности» или «рид секвенирования нуклеиновой кислоты» обозначает любую информацию или данные, которые указывают на порядок нуклеотидных оснований (таких как аденин, гуанин, цитозин и тимин/урацил) в молекуле (например, целого генома, целого транскрипта, экзома, олигонуклеотида, полинуклеотида, фрагмента и т.д.) ДНК или РНК.
Необходимо понимать, что, согласно идеям настоящего изобретения, рассматривается информация последовательности, полученная с использованием всего доступного разнообразия способов, платформ или технологий, включая, но не ограничиваясь указанными: капиллярный электрофорез, микрочипы, системы на основе лигирования, системы на основе полимеразы, системы на основе гибридизации, системы прямой или непрямой идентификации нуклеотида, пиросеквенирования, системы делеции на основе ионов или рН, системы на основе электронной подписи и т.д.
Ссылка на основание, нуклеотид или другую молекулу может относиться к единственному или множественному числу. То есть «основание» может относиться к одной молекуле указанного основания или ко множеству оснований, например, в растворе.
Термин «полинуклеотид», «нуклеиновая кислота» или «олигонуклеотид» относится к линейному полимеру нуклеозидов (включая дезоксирибонуклеозды, рибонуклеозды или их аналоги), соединенных внутринуклеозидными связями. Как правило, полинуклеотид содержит по меньшей мере три нуклеозида. Как правило, размер олигонуклеотидов варьирует от нескольких мономерных единиц, например 3-4, до нескольких сотен мономерных единиц. Во всех случаях, когда полинуклеотид, такой как олигонуклеотид, представлен буквами последовательности, такими как «ATGCCTG», необходимо понимать, что нуклеотиды расположены в порядке от 5' к 3' слева направо, и что «А» означает дезоксиаденозин, «С» означает дезоксицитидин, «G» означает дезоксигуанозин и «Т» означает тимидин, если иное не указано. Буквы А, С, G и Τ могут использоваться для ссылки на сами основания, на нуклеозиды или на нуклеотиды, содержащие указанные основания, что является стандартным в данной области техники.
При использовании в настоящей заявке, термин «нуклеиновая кислота-мишень» или «нуклеотидная последовательность-мишень» относится к любой нуклеотидной последовательности (например, РНК или ДНК), проведение манипуляций с которой специалист в данной области техники может считать желаемыми по какой-либо причине. В некоторых контекстах «нуклеиновая кислота-мишень» относится к нуклеотидной последовательности, нуклеотидная последовательность которой подлежит определению или нуклеотидную последовательность которой желательно определить. В некоторых контекстах термин «нуклеотидная последовательность-мишень» относится к последовательности, к которой создан частично или полностью комплементарный праймер или зонд.
При использовании в настоящей заявке, термин «интересующая область» относится к нуклеиновой кислоте, которую анализируют (например, с использованием одной из композиций, систем или способов, описанных в настоящей заявке). Согласно некоторым вариантам реализации изобретения, интересующая область представляет собой часть генома или участок геномной ДНК (например, содержащий одну или более хромосом или один или более генов). Согласно некоторым вариантам реализации изобретения, анализируют мРНК, которая экспрессируется с интересующей области.
При использовании в настоящей заявке термин «соответствует» или «соответствующий» используется для ссылки на непрерывную последовательность нуклеиновой кислоты или нуклеотидную последовательность (например, субпоследовательность), которая комплементарна, и, таким образом, «соответствует», всей или части последовательности нуклеиновой кислоты-мишени.
При использовании в настоящей заявке фраза «клонированное множество нуклеиновых кислот» относится к продуктам нуклеиновой кислоты, которые представляют собой полные или частичные копии матрицы нуклеиновой кислоты, на основе которой они были созданы. Указанные продукты по существу или полностью или фактически идентичны друг другу и представляют собой комплементарные копии цепи матрицы нуклеиновой кислоты, с которой они синтезируются, при условии что частота ошибок включения нуклеотида во время синтеза клональных молекул нуклеиновой кислоты составляет 0%.
При использовании в настоящей заявке, термин «библиотеки» относится ко множеству нуклеиновые кислоты, например, множеству разных нуклеиновых кислот.
При использовании в настоящей заявке, «субпоследовательность» нуклеотидной последовательность относится к любой нуклеотидной последовательности, содержащейся в пределах нуклеотидной последовательности, включая любую субпоследовательность размером от одного основания до размера, на одно основание меньшего нуклеотидной последовательности.
При использовании в настоящей заявке, термин «консенсусная последовательность» относится к последовательности, которая является общей или по другой причине присутсвуют в самой большой фракции группы выровненных последовательностей. Консенсусная последовательность описывает нуклеотиды, наиболее часто встречающиеся в каждом положении в пределах групп последовательностей нуклеиновых кислот. Консенсусная последовательность часто «собирается» из более коротких прочтений последовательности.
При использовании в настоящей заявке, «сборка» относится к созданию информации нуклеотидной последовательности из более коротких последовательностей, например, экспериментально полученных прочтений последовательности. Сборку последовательности в целом можно разделить на две большие категории: сборка de novo и сборка картированием к референсному геному. При сборке de novo прочтения последовательности собираются вместе таким образом, что они образуют новую и ранее не известную последовательность. При «картировании» к референсному геному прочтения последовательности собираются относительно существующей «референсной последовательности» для создания последовательности, которая подобна, но не обязательно идентична указанной рефернсной последовательности.
Фраза «рабочий цикл секвенирования» относится к любому этапу или части эксперимента по секвенированию, осуществляемому для определения некоторой информации относительно по меньшей мере одной биомолекулы (например, молекулы нуклеиновой кислоты).
При использовании в настоящей заявке фраза «dNTP» означает дезоксинуклеотидтрифосфат, где нуклеотид содержит нуклеотидное основание, такое как А, Т, С, G или U.
Термин «мономер» при использовании в настоящей заявке означает любое соединение, которое может быть включено в растущую молекулярную цепь с помощью конкретной полимеразы. Такие мономеры включают, но не ограничиваются указанными, природные нуклеотиды (например, ATP, GTP, ТТР, UTP, СТР, dATP, dGTP, dTTP, dUTP, dCTP, синтетические аналоги), предшественники каждого нуклеотида, неприродные нуклеотиды и их предшественники или любую другую молекулу, которая может быть включена в растущую цепь полимера с помощью данной полимеразы.
При использовании в настоящей заявке термин «комплементарный» в целом относится к конкретному дуплексу нуклеотидов с образованием канонических пар оснований Уотсона-Крика, как очевидно специалистам в данной области техники. Однако термин «комплементарный» также включает спаривание оснований нуклеотидных аналогов, которые способны к универсальному спариванию с нуклеотидами А, Т, G или С, и запертыми нуклеиновыми кислотами, которые усиливают термическую стабильность дуплексов. Специалист в данной области техники определит, что точность гибридизации представляет собой детерминанту степени совпадения или отсутствия совпадения в дуплексе, образованном в результате гибридизации.
«Полимераза» представляет собой фермент, в целом предназначенный для соединения 3'-ОН 5'-трифосфатнуклеотидов, олигомеров и их аналогов. Полимеразы включают, но не ограничиваются указанными, ДНК-зависимые ДНК-полимеразы, ДНК-зависимые РНК-полимеразы, РНК-зависимые ДНК-полимеразы, РНК-зависимые РНК-полимеразы, ДНК-полимеразу Т7, ДНК-полимеразу Т3, ДНК-полимеразу Т4, РНК-полимеразу Т7, РНК-полимеразу Т3, РНК-полимеразу SP6, ДНК-полимеразу 1, фрагмент Кленова, ДНК-полимеразу Thermophilus aquaticus (Taq), ДНК-полимеразу Thermus thermophilus (Tth), ДНК-полимеразу Vent (New England Biolabs), ДНК-полимеразу Deep Vent (New England Biolabs), ДНК-полимеразу, большой фрагмент ДНК-полимеразы Bacillus stearothermophilus (Bst), фрагмент Стефеля, ДНК-полимеразу 9°N, полимеразу 9°Nm, ДНК-полимеразу Pyrococcus furiosis (Pfu), ДНК-полимеразу Thermus filiformis (Tfl), полимеразу RepliPHI Phi29, ДНК-полимеразу Thermococcus litoralis (Tli), эукариотическую ДНК-полимеразу бета, теломеразу, полимеразу Therminator (например, THERMINATOR I, THERMINATOR II и т.д., New England Biolabs), ДНК-полимеразу KOD HiFi. (Novagen), ДНК-полимеразу KOD1, Q-бета репликазу, концевую трансферазу, отбратную траснкриптазу AMV, обратную траснкриптазу M-MLV, обратную траснкриптазу Phi6, отбратную траснкриптазу ВИЧ-1, новые полимеразы, открытые в процессе биоразработок и/или молекулярной эволюции, и полимеразы, цитированные в публикации заявки на патент США №2007/0048748 и в патентах США №6,329,178; 6,602,695 и 6,395,524. Указанные полимеразы включают полимеразы дикого типа, мутантные изоформы и генетически сконструированные варианты, такие как экзополимеразы; полимеразы с минимальной, не поддающейся выявлению и/или пониженной 3'→5' коррекционной экзонуклеазной активностью и другие мутанты, например, которые принимающие меченые нуклеотиды и встраивающие их в цепь нуклеиновой кислоты. Согласно некоторым вариантам реализации изобретения, полимераза создана для применения, например, в ПЦР в реальном времени, ПЦР с высокой точностью воспроизведения, ДНК-секвенирования нового поколения, быстрой ПЦР, ПЦР с горячим стартом, ПЦР неочищенного образца, глубокой ПЦР и/или молекулярной диагностики. Такие ферменты доступны от многих коммерческих поставщиков, например, Kapa Ferments, Finnzymes, Promega, Invitrogen, Life Technologies, Thermo Scientific, Qiagen, Roche и т.д.
Термин «праймер» относится к олигонуклеотиду, который встречается в природе в виде очищенного фрагмента рестрикции или получен синтетическим путем, который способен служить точкой инициации синтеза при помещении в условия, в которых инициируется синтез продукта удлинения праймера, который комплементарен цепи нуклеиновой кислот (например, в присутствии нуклеотидов и индуцирующего агента, такого как ДНК-полимераза, и при подходящей температуре и рН). Праймер предпочтительно является одноцепочечным для максимальной эффективности в амплификации, но может, альтернативно, являться двуцепочечным. Если праймер является двуцепочечным, он сначала обрабатывается для разделения его цепей до его использования для получения продуктов удлинения. Предпочтительно, праймер представляет собой олигодезоксирибонуклеотид. Праймер должен быть достаточно длинным для праймирования синтеза продуктов удлинения в присутствии индуцирующего агента. Точная длина праймеров зависит от многих факторов, включая температуру, источник праймера и способ применения.
При использовании в настоящей заявке, «адаптер» представляет собой олигонуклеотид, который связан или создан для связывания с нуклеиновой кислотой для введения указанной нуклеиновой кислоты в производственный процесс секвенирования. Адаптер может быть одноцепочечным или двуцепочечным (например, двуцепочечная ДНК или одноцепочечная ДНК). При использовании в настоящей заявке, термин «адаптер» относится к адаптерной нуклеиновой кислоте в состоянии, не связанном с другой нуклеиновой кислотой и в состоянии, связанном с другой нуклеиновой кислотой.
По меньшей мере часть адаптера содержит известную последовательность. Например, некоторые варианты реализации адаптеров включают связывающуюся с праймером последовательность для амплификации нуклеиновой кислоты и/или для связывания праймера для секвенирования. Некоторые адаптеры включают последовательность для гибридизации комплементарной зонда захвата. Некоторые адаптеры включают химический или другой фрагмент (например, фрагмент биотина) для захвата и/или иммобилизации на твердом носителе (например, содержащем авидиновый фрагмент). Некоторые варианты реализации адаптеров включают маркер, индекс, штрихкод, метку или другую последовательность, с помощью которой адаптер и нуклеиновая кислота, к которой он присоединен, выявляются.
Некоторые адаптеры включают универсальную последовательность. Универсальная последовательность представляет собой последовательность, общую для множества адаптеров, последовательности которых иметь другие отличия вне универсальной последовательности. Например, универсальная последовательность обеспечивает общий сайт связывания праймера для сборки нуклеиновых кислот на основе различных нуклеиновых кислот-мишеней, которые, например, могут содержать разные штрихкоды.
Некоторые варианты реализации адаптеров имеют определенную, но неизвестную последовательность. Например, некоторые варианты реализации адаптеров включают вырожденную последовательность с определенным числом оснований (например, вырожденную последовательность, содержащую от 1 до 20 оснований). Такая последовательность является определенной, даже если каждая отдельная последовательность неизвестна; такая последовательность тем не менее может служить в качестве индекса, штрихкода, метки и т.д., маркируя фрагменты нуклеиновой кислоты, например, одной и той же нуклеиновой кислоты-мишени.
Некоторые адаптеры имеют тупой конец, и некоторые адаптеры имеют «липкий» конец из одного или более оснований.
Согласно конкретным вариантам реализации изобретения, предложенные в настоящей заявке адаптеры содержат фрагмент азидо, например, адаптер содержит азидо (например, азидометил) фрагмент на 5' конце. Таким образом, некоторые варианты реализации относятся к адаптерам, которые представляют собой или включают 5'-азидо-модифицированный олигонуклеотид или 5'-азидометил-модифицированный олигонуклеотид.
При использовании в настоящей заявке, «система» означает набор компонентов, реальных или абстрактных, составляющих целое, где каждый указанный компонент взаимодействует или относится по меньшей мере к одному другому компоненту в пределах целого.
При использовании в настоящей заявке «индекс» в целом означает отличительную или идентификационную черту или характеристику. Один пример индекса представляет собой короткую нуклеотидную последовательность, используемую в качестве «штрихкода» для идентификации более длинного нуклеотида, содержащего штрихкод и другую последовательность.
При использовании в настоящей заявке термин «фаза» или «фазирование» относится к уникальному содержимому двух хромосом, наследуемому от каждого родителя и/или дифференцирующему информацию последовательности, присутствующей в нуклеиновой кислоте (например, хромосоме), произошедшую от матери и от отца. Например, фазирование информации гаплотипа описывает, какие нуклеотиды (например, SNP), области, части или фрагменты произошли от каждой из родительских хромосом (или связаны со специфичным минорным вирусным квазивидом).
При использовании в настоящей заявке «лестница» Сэнджера, «лестница» ДНК», «лестница» фрагментов или «лестница» относится к библиотеке нуклеиновых кислот (например, ДНК), все из которых отличаются в длину на небольшое число оснований, например, от одного до пяти оснований, и в некоторых предпочтительных вариантах реализации на одно основание. Согласно некоторым вариантам реализации изобретения, 5' концы нуклеиновых кислот в «лестнице» соответствуют тем же положениям нуклеотидов (или находятся в пределах небольшого диапазона положений нуклеотидов, например, 1-10 положений нуклеотидов) в матрице, на основе которой они были созданы, и 3' концы указанных нуклеиновых кислот в «лестнице» являются разными и соответствуют диапазону положений нуклеотидов в матрице, на основе которой они были созданы. См., например, типичные «лестницы» и/или «лестницы», подобные предложенным в настоящей заявке в источнике Sanger & Coulson (1975) "A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase" J Mol Biol 94(3): 441-8," Sanger et al (1977) "DNA sequencing with chain-terminating inhibitors" Proc Natl Acad Sci USA 74 (12): 5463-7.
Описание
Согласно некоторым вариантам реализации изобретения, технология, предложенная в настоящей заявке, обеспечивает способы и композиции для получения коротких перекрывающиеся фрагментов ДНК, которые покрывают более длинную область фрагмента ДНК. В частности, короткие фрагменты ДНК составляют популяцию фрагментов ДНК, размер которых различается и повышается в порядке от одного фрагмента к следующему более большому фрагменту, например, на 1-20 пар оснований, 1-10 пар оснований или 1-5 пар оснований, предпочтительно на 1 пару оснований (например, как в случае фрагментов, полученных в результате секвенирования по Сэнджеру). Согласно некоторым вариантам реализации изобретения, короткая нуклеиновая кислота, имеющая универсальную последовательность, присоединена к 3' концу каждого фрагмента (например, концу фрагмента, по которому строится «лестница»). Затем фрагменты секвенируют с использованием праймера секвенирования, комплементарного универсальной последовательности. Таким образом, полученные последовательности имеют диапазон 5' (первых) оснований, соответствующих основаниям, распределенным вдоль более длинной ДНК от первого основания, присоединенного к универсальной последовательности, до 500 оснований или более. Предпочтительно, полученные последовательности имеют диапазон 5' (первых) оснований, соответствующих каждому основанию, распределенному вдоль более длинной ДНК. С помощью указанного способа короткие прочтения NGS (от ~30 до ~50 оснований) используются для сборки длинного непрерывного прочтения, который сохраняет информацию фазирования и/или сцепления (см., например, Фигуру 1).
1. Способы получения библиотек для NGS
Варианты реализации технологии изображены с помощью схемы, показанной на Фигуре 2. Во-первых, согласно некоторым вариантам реализации изобретения, нуклеиновую кислоту-мишень амплифицируют с использованием одного или более мишень-специфичных праймеров (см., например, Фигуру 2А, этап i; Фигуру 2С, этап i). Нуклеиновая кислота-мишень может представлять собой ДНК или РНК, например, геномную ДНК; мРНК; космиду, фосмиду или бактериальные искусственные хромосомы (например, содержащие инсерцию), ген, плазмиду и т.д. Согласно некоторым вариантам реализации изобретения, РНК сначала подвергается обратной транскрипции с получением ДНК. Амплификация может представлять собой ПЦР, ПЦР с ограниченным числом циклов (с малым количеством циклов, например, 5-15 циклами (например, 8 циклами)), изотермическую ПЦР, амплификацию с ферментами Phi29 или Bst и т.д., например, как показано на Фигуре 2А и на Фигуре 2С.
Согласно некоторым вариантам реализации изобретения, мишень-специфичные праймеры включают как универсальную последовательность (например, универсальную последовательность А), так и уникально идентифицируемую индексную последовательность (например, штрихкод-последовательность; см. Фигуру 2А, «NNNNN» штрихкод-последовательность), которая позволяет трэкинг и/или идентификацию нуклеиновой кислоты-мишени, из которой был получен амплифицированный продукт (ампликон). В целом, штрихкод-последовательности могут состоять из 1 до 10 или более нуклеотидов. Например, штрихкод-последовательность из 10 оснований обеспечивает 1,048,576 (410) комбинаций уникально поддающихся идентификации молекул специфичного по отношению к мишени праймера. В результате при созданной подходящим образом длине штрихкода исходный материал, содержащий от небольшого до очень большого числа фрагментов ДНК-мишеней, можно легко метить и индексировать без повторного мечения той же штрихкод последовательностью.
Согласно некоторым вариантам реализации изобретения, праймеры используются для амплификации (например, не включают штрихкод), и ампликон-мишень лигируется с адаптером, который содержит одну или более универсальных последовательностей и/или одну или более штрихкод последовательностей (см., например, Фигуру 2С, «NNNNNNNNNN» штрихкод-последовательность, этап ii). Таким образом, согласно некоторым вариантам реализации изобретения, следующий этап включает лигирование адаптера с ампликоном-мишенью. Согласно некоторым вариантам реализации изобретения, адаптер содержит первую цепь, содержащую вырожденную последовательность (например, содержащую от 8 до 12 оснований), фланкированную как на 5' конце, так и на 3' конце двумя разными универсальными последовательностями (например, универсальной последовательностью А и универсальной последовательности В; см. Фигуру 9), и вторую цепь, содержащую универсальную последовательность С (например, на 5' конце), и последовательность (например, на 3' конце), которая комплементарна универсальной последовательности В и которая содержит дополнительный Τ в 3'-концевом положении.
В настоящей заявке предложены варианты реализации изобретения для получения «лестницы» фрагментов из циркуляризованной матрицы (см., например, Фигуру 2А и Фигуру 2В) и варианты реализации изобретения для получения «лестницы» фрагментов из линейной матрицы (см., например, Фигуру 2С). Соответственно, согласно некоторым вариантам реализации изобретения, следующий этап включает лигирование 3' концов уникально штрихкодированных отдельных ампликонов с адаптерным олигонуклеотидом, составляющим приблизительно от 10 до 80 оснований в длину и содержащим вторую универсальную последовательность (например, универсальную последовательность В) (см., например, Фигуру 2А, этап ii). После лигирования нуклеиновые кислоты адаптер-ампликон самолигируются (например, подвергаются циркуляризаци) с получением кольцевой матрицы (см., например, Фигуру 2А, этап iii). Циркуляризация приводит к смежному расположению 3' конца универсальной последовательности с 5' концом штрихкод-последовательности. Внутримолекулярное лигирование можно осуществлять с использованием лигазы. Например, CircLigase II (Epicentre) представляет собой термостабильную одноцепочечную ДНК-лигазу, которая катализирует внутримолекулярное лигирование одноцепочечных ДНК-матриц, содержащих 5' фосфатную и 3' гидроксильную группу.
Кроме того, согласно вариантам реализации, связанным с использованием циркуляризованной матрицы, «лестница» ДНК по типу фрагментов Сэнджера создана с помощью полимеразной реакции с использованием праймера, комплементарного универсальной последовательности А, и смеси dNTP и 3'-O-блокированных dNTP аналогов, как описано в настоящей заявке (см, например, Фигуру 2А, этап iv). Согласно некоторым вариантам реализации изобретения, 3'-О-заблокированный аналог dNTP представляет собой 3'-О-алкинил-нуклеотидный аналог (например, алкил, имеющий насыщенное положение (sp3-гибридизованное) на молекулярной рамке, следующей за алкинильной группой, и их замещенные варианты). Согласно некоторым вариантам реализации изобретения, 3'-О-заблокированный аналог dNTP представляет собой 3'-O-пропаргил-нуклеотидный аналог, имеющий структуру, как показано ниже:

где В представляет собой основание нуклеотида (например, аденин, гуанин, тимин, цитозин или природное или синтетическое нуклеиновое основание, например, модифицированный пурин, такой как гипоксантин, ксантин, 7-метилгуанин; модифицированный пиримидин, такой как 5,6-дигидроурацил, 5-метилцитозин, 5-гидроксиметилцитозин и т.д.), и Ρ содержит фосфатный фрагмент. Согласно некоторым вариантам реализации изобретения, Ρ содержит тетрафосфат; трифосфат; дифосфат; монофосфат; 5' гидроксил; альфа-тиофосфат (например, фосфоротиоат или фосфородитиоат), бета-тиофосфат (например, фосфоротиоат или фосфородитиоат), и/или гамма-тиофосфат (например, фосфоротиоат или фосфородитиоат) или -альфа метилфосфонат, бета-метилфосфонат и/или гамма-метилфосфонат. Другие алкинильные группы рассматриваются и применяются согласно настоящей технологии, например, бутинил и т.д. Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог является таким, как описано в других разделах в настоящей заявке.
Альтернативно, согласно вариантам реализации, связанным с применением линейной матрицы (см., например, Фигуру 2С), «лестница» ДНК по типу фрагментов Сэнджера создается с помощью полимеразной реакции с использованием праймера, комплементарного последовательности в адаптере, и смеси dNTP и 3'-O-блокированных аналогов dNTP, как описано в настоящей заявке (см., например, Фигуру 2С, этап iii). Согласно некоторым вариантам реализации изобретения, 3'-O-блокированный аналог dNTP представляет собой 3'-О-алкинил-нуклеотидный аналог (например, алкил, имеющий насыщенное положение (sp3-гибридизованное) на молекулярной рамке, следующей за алкинильной группой, и их замещенные варианты). Согласно некоторым вариантам реализации изобретения, 3'-O-блокированный аналог dNTP представляет собой 3'-O-пропаргил-нуклеотидный аналог, имеющий структуру, показанную ниже:
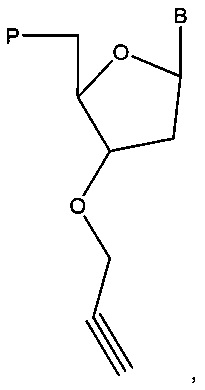
где В представляет собой основание нуклеотида (например, аденин, гуанин, тимин, цитозин или природное или синтетические нуклеиновое основание, например, модифицированный пурин, такой как гипоксантин, ксантин, 7-метилгуанин; модифицированный пиримидин, такой как 5,6-дигидроурацил, 5-метилцитозин, 5-гидроксиметилцитозин; и т.д.), и Р содержит фосфатный фрагмент. Согласно некоторым вариантам реализации изобретения, Р содержит тетрафосфат; трифосфат; дифосфат; монофосфат; 5' гидроксил; альфатиофосфат (например, фосфоротиоат или фосфородитиоат), бета-тиофосфат (например, фосфоротиоат или фосфородитиоат) и/или гамма-тиофосфат (например, фосфоротиоат или фосфородитиоат) или альфа-метилфосфонат, бета-метилфосфонат и/или гамма-метилфосфонат. Другие алкинильные группы рассматриваются и применяются согласно настоящей технологии, например, бутинил и т.д. Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог является таким, как описано в других разделах в настоящей заявке.
Варианты реализации технологии обеспечивают преимущества по сравнению с существующими технологиями. Например, согласно некоторым вариантам реализации изобретения, технология обеспечивает последовательность высокого качества из малого количества исходной нуклеиновой кислоты (например, менее чем 10 нг нуклеиновой кислоты, например, менее чем 10 нг геномной ДНК). Технология обеспечивает стабильное мечение отдельных матриц. Создание библиотек является эффективным, поскольку способы включают мало манипуляций (и, таким образом, мало этапов очистки), и каждая из манипуляций приводит к достаточному выходу.
Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог содержит обратимый терминатор, который содержит блокирующую группу, которую можно удалить для разблокирования нуклеотида. Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог содержит функциональный терминатор, например, который обеспечивает конкретную желаемую реактивность для следующих далее групп.
Нуклеотидные аналоги приводят к получению «лестницы» фрагментов, содержащей фрагменты в диапазоне размеров. Например, согласно некоторым вариантам реализации изобретения, длина фрагментов варьирует от приблизительно 10 до приблизительно 50 п.о., от приблизительно 10 до приблизительно 100 п.о., и до приблизительно 100 п.о. до приблизительно 700 или приблизительно 800 п.о. или более п.о.; более того, согласно некоторым вариантам реализации изобретения, длину более 1000 п.о. можно достигать путем регулирования соотношения dNTP и 3'-О-блокированных аналогов dNTP в реакционной смеси (например, путем использования соотношения, составляющего от 1:500 до 500:1 (например, 1:500, 1:450, 1:400, 1:350, 1:300, 1:250, 1:200, 1:150, 1:100, 1:90, 1:80, 1:70, 1:60, 1:50, 1:40, 1:30, 1:20, 1:10, 1:9, 1:8, 1:7, 1:6, 1:5, 1:4, 1:3, 1:2, 2:1, 3:1, 4:1, 5:1, 6:1, 7:1, 8:1, 9:1, 10:1, 20:1, 30:1, 40:1, 50:1, 60:1, 70:1, 80:1, 90:1, 100:1, 150:1, 200:1, 250:1, 300:1, 350:1, 400:1, 450:1 или 500:1).
Стандартные технологии секвенирования на основе дидезоксинуклеотидов (ddNTP) (например, химические реакции секвенирования по типу Сэнджера) не подходят для данного этапа в указанных вариантах реализации, поскольку отсутствие 3'-ОН группы в терминирующем ddNTP приводит к получению нереакционноспособного 3' конца, который не может участвовать в лигировании второго адаптерного олигонуклеотида в последующем этапе.
Когда получена «лестница» фрагментов нуклеиновой кислоты с реакционноспособными (например, подающимися лигированию) 3' концами, второй адаптерный олигонуклеотид, содержащий универсальную последовательность (например, универсальную последовательность С) лигируют (ферментативно или химически) с 3' концами фрагментов «лестницы» фрагментов нуклеиновой кислоты с получением библиотеки NGS. (см., например, Фигуру 2А, этап v; Фигуру 2С, этап (iv)). Согласно некоторым вариантам реализации изобретения, для амплификации конечного продукта осуществляют ПЦР с ограниченным количеством циклов или другой способ амплификации.
Согласно некоторым вариантам реализации изобретения, способы применяются в получении коротких последовательностей, например, состоящих из ~120-200 п.о. Такие варианты реализации применяются, например, в анализе раковых генов, например, для анализа мутаций панели раковых заболеваний. Согласно некоторым вариантам реализации изобретения, технология применяется для получения последовательностей, состоящих из 500 п.о., 1000 п.о. или более. Например, согласно некоторым вариантам реализации изобретения, нуклеиновую кислоту-мишень амплифицируют с использованием одного или более специфичных по отношению к мишени праймеров (см., например, Фигуру 2В, этап i; Фигуру 2С, этап (i)). Нуклеиновая кислота-мишень может представлять собой ДНК или РНК, например, геномную ДНК; мРНК; космиду, фосмиду или бактериальные искусственные хромосомы (например, содержащие инсерции), ген, плазмиду и т.д. Согласно некоторым вариантам реализации изобретения, РНК сначала подвергается обратной транскрипции с получением ДНК. Реакция амплификации может представлять собой ПЦР, ПЦР с ограниченным количеством циклов, изотермическую ПЦР, амплификацию с помощью ферментов Phi29 или Bst и т.д., например, как показано на Фигуре 2В и на Фигуре 2С.
Согласно некоторым вариантам реализации изобретения, мишень-специфичные праймеры включают как универсальную последовательность (например, универсальную последовательность А), так и уникально идентифицируемую индексную последовательность (например, штрихкод последовательность; см. Фигуру 2В, «NNNNN» штрихкод последовательность), которая позволяет трэкинг и/или идентификацию нуклеиновой кислоты-мишени, на основе которой был получен амплифицированный продукт (ампликон). В целом, штрихкод-последовательности могут содержать от 1 до 10 или более нуклеотидов. Например, штрихкод-последовательность из 10 оснований обеспечивает 1,048,576 (410) комбинаций уникально поддающихся идентификации молекул специфичного по отношению к мишени праймера. В результате при созданной подходящим образом длине штрихкода исходный материал, содержащий от небольшого до очень большого числа фрагментов ДНК-мишеней, можно легко метить и индексировать без повторного мечения той же штрихкод-последовательностью.
Согласно некоторым вариантам реализации изобретения, следующий этап включает лигирование 3' концов уникально штрихкодированных отдельных ампликонов с адаптерным олигонуклеотидом, составляющим приблизительно от 10 до 80 оснований в длину и содержащим вторую универсальную последовательность (например, универсальную последовательность В) (см., например, Фигуру 2В, этап ii). После лигирования нуклеиновые кислоты адаптер-ампликон самолигируются (например, подвергаются циркуляризации) с образованием кольцевой матрицы (см., например, Фигуру 2В, этап iii). Циркуляризация приводит к смежному расположению 3' конца универсальной последовательности с 5' концом штрихкод-последовательности. Внутримолекулярное лигирование можно осуществлять с использованием лигазы. Например, CircLigase II (Epicentre) представляет собой термостабильную одноцепочечную ДНК лигазу, которая катализирует внутримолекулярное лигирование одноцепочечной ДНК матрицы, содержащей 5' фосфатную и 3' гидроксильную группу.
С использованием циркуляризованной матрицы «лестницам ДНК по типу фрагментов Сэнджера создается с помощью полимеразной реакции с использованием праймера, комплементарного универсальной последовательности А и смеси dNTP и 3'-О-блокированных аналогов dNTP, как описано в настоящей заявке (см., например, Фигуру 2В, этап iv). Согласно некоторым вариантам реализации изобретения, 3'-O-блокированных аналог dNTP представляет собой 3'-O-алкинил-нуклеотидный аналог (например, алкил, имеющий насыщенное положение (sp3-гибридизованное) на молекулярной рамке, следующей за алкинильной группой, и их замещенные варианты). Согласно некоторым вариантам реализации изобретения, 3'-O-блокированный аналог dNTP представляет собой 3'-О-пропаргил-нуклеотидный аналог, имеющий структуру, показанную ниже:
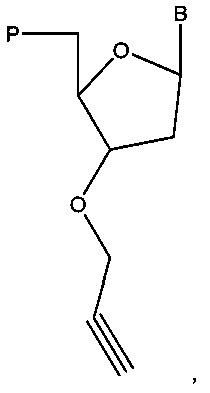
где В представляет собой основание нуклеотида (например, аденин, гуанин, тимин, цитозин или природное или синтетические нуклеиновое основание, например, модифицированный пурин, такой как гипоксантин, ксантин, 7-метилгуанин; модифицированный пиримидин, такой как 5,6-дигидроурацил, 5-метилцитозин, 5-гидроксиметилцитозини т.д.), и Ρ содержит фосфатный фрагмент. Согласно некоторым вариантам реализации изобретения, Ρ содержит тетрафосфат; трифосфат; дифосфат; монофосфат; 5' гидроксил; альфа-тиофосфат (например, фосфоротиоат или фосфородитиоат), бета-тиофосфат (например, фосфоротиоат или фосфородитиоат), и/или гамма-тиофосфат (например, фосфоротиоат или фосфородитиоат); или альфа-метилфосфонат, бета-метилфосфонат и/или гамма-метилфосфонат. Другие алкинильные группы рассматриваются и применяются согласно технологии, например, бутинил и т.д. Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог является таким, как описано в других разделах в настоящей заявке. Другие алкинильные группы рассматриваются и применяются согласно технологии, например, бутинил и т.д. Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог является таким, как описано в других разделах в настоящей заявке.
Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог содержит обратимый терминатор, который содержит блокирующую группу, которая может быть удалена для разблокировки нуклеотида. Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог содержит функциональный терминатор, например, который обеспечивает конкретную желаемую реакционную способность для последующих этапов. Нуклеотидные аналоги приводят к получению «лестницы» фрагментов, содержащей фрагменты в диапазоне размеров. Например, согласно некоторым вариантам реализации изобретения, фрагменты имеют длину, варьирующую от ~100 п.о. до ~700 или 800 п.о.; более того, согласно некоторым вариантам реализации изобретения, длину последовательности, составляющую от более чем 1000 п.о. до более чем 10,000 п.о., можно достигать, например, путем регулирования соотношения dNTP и 3'-O-блокированных dNTP аналогов в реакционной смеси.
Стандартные технологии секвенирования на основе дидезоксинуклеотидов (ddNTP) (например, химические реакции секвенирования по типу Сэнджера) не подходят для данного этапа в указанных вариантах реализации изобретения, поскольку отсутствие 3'-ОН группы в терминирующем ddNTP приводит к получению нереакционноспособного 3' конца, который не может участвовать в лигировании второго адаптерного олигонуклеотида в последующем этапе.
Кроме того, «лестница» фрагментов нуклеиновой кислоты циркуляризована для образования библиотеки циклических нуклеиновых кислот (см., например, Фигуру 2В, этап ν). После переваривания с помощью одного или более рестрикционных ферментов (см., например, Фигуру 2В, этап vi) второй адаптерный олигонуклеотид (например, содержащий универсальную последовательность, например, универсальную последовательность С) лигируют (ферментативно или химически) с 3' концами продуктов переваривания библиотеки циклических нуклеиновых кислот с получением библиотеки NGS. (см., например, Фигуру 2В, этап vii). Согласно некоторым вариантам реализации изобретения, для амплификации конечного продукта осуществляют ПЦР с ограниченным количеством циклов или другой способ амплификации. Без ограничения каким-либо конкретным способом или временем осуществления каких-либо этапов описанных способов, осуществление способов, предложенных согласно некоторым вариантам реализации изобретения, занимает от ~6 (например, -6,5) часов до ~9 (например, ~8,5 часов).
Согласно некоторым вариантам реализации изобретения (например, вариантам реализации с использованием терминаторов, представляющих собой 3'-О-алкинил-нуклеотидные аналог, такие как 3'-О-пропаргил-нуклеотидные аналоги), фрагменты включают 3' алкин. Кроме того, согласно некоторым вариантам реализации изобретения, второй адаптерный олигонуклеотид, содержащий универсальную последовательность (например, универсальную последовательность С) содержит 5' азидную группу (N3), которая может реагировать с фрагментом 3' алкиновой группы. Кроме того, согласно некоторым вариантам реализации изобретения, процесс «клик-химии», такой как азид-алкиновое циклоприсоединение, используется для связывания адаптера с фрагментом через образование триазола:
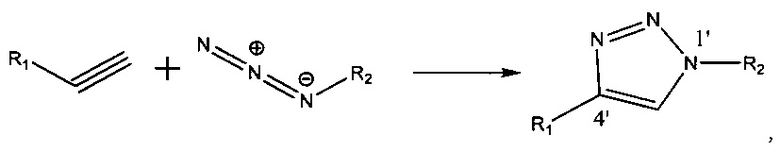
где R1 и R2 по отдельности представляют собой любую химическую структуру или химический фрагмент.
Согласно некоторым вариантам реализации изобретения, связь триазольного кольца имеет структуру в соответствии со следующими структурами:
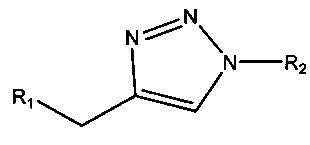 , например,
, например, 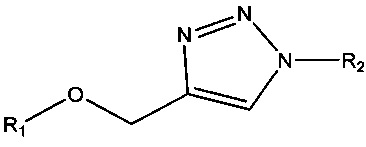 , например,
, например,
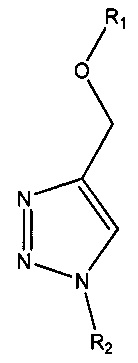 , например,
, например, 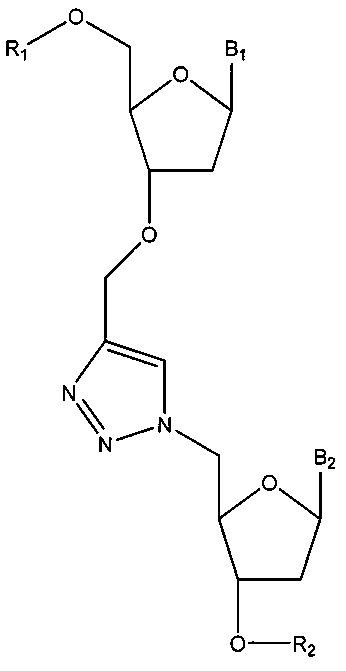 ,
,
где R1 и R2 по отдельности представляют собой любую химическую структуру или химический фрагмент (не обязательно одинаковый для разных структур), и В, B1, и В2 по отдельности обозначают основание нуклеотида (например, аденин, гуанин, тимин, цитозин или природное или синтетические нуклеиновое основание, например, модифицированный пурин, такой как гипоксантин, ксантин, 7-метилгуанин; модифицированный пиримидин, такой как 5,6-дигидроурацил, 5-метилцитозин, 5-гидроксиметилцитозини т.д.).
Связь триазольного кольца, образованная в результате азид алкинового циклоприсоединения, имеет сходные характеристики (например, физические, биологические, химические характеристики) с природной фосфодиэфирной связью, присутствующей в нуклеиновых кислотах, и таким образом, имитирует скелет нуклеиновой кислоты. В результате стандартные ферменты, которые распознают природные нуклеиновые кислоты в качестве субстратов, также распознают в качестве субстратов продукты, образованные путем азид-алкинового циклоприсоединения, как предложено согласно технологии, описанной в настоящей заявке. См., например, источник El-Sagheer, et al. (2011) "Biocompatible artificial DNA linker that is read through by DNA polymerases and is functional in Escherichia coli" Proc Natl Acad Sci USA 108(28): 11338-43, полностью включенный в настоящую заявку посредством ссылки).
Конечную библиотеку фрагментов NGS затем используют в качестве входных данных для системы секвенирования NGS. Во время секвенирования секвенируют от ~20 до 50 оснований ДНК, расположенной рядом с адаптером, содержащим универсальную последовательность С (соответствующую ~20-50 основаниям нуклеиновой кислоты-мишени) и штрихкод, смежный с адаптером, содержащим универсальную последовательность В (см., например, Фигуру 3). Когда последовательности получены, прочтения последовательностей распределяют в группы на основании штрихкод-последовательностей для сбора прочтений последовательности, которая происходит из молекулы матрицы, меченной указанной конкретной уникальной штрихкод-последовательностью (см., например, Фигуру 3). Прочтения последовательностей в каждой группе (для каждой штрихкод-последовательности) выравнивают относительно друг друга и собирают для конструирования более длинной непрерывной консенсусной последовательности с ненарушенной информацией фазирования. Указанная последовательность может быть выровнена по отношению к соответствующей референсной последовательности для последующего анализа последовательности.
Различные примеры платформ для секвенирования нуклеиновых кислот, систем сборки нуклеиновых кислот и/или картирования нуклеиновых кислот (например, компьютерные программы и/или оборудование) описаны, например, в публикации заявки на патент США №2011/0270533, которая включена в настоящую заявку посредством ссылки. Технологии секвенирования «спаренных концов», «партнеров» и другие связанные со сборкой способы секвенирования в целом известны в области молекулярной биологии (Siegel A.F. et al., Genomics 2000, 68: 237-246; Roach J. С.et al., Genomics 1995, 26: 345-353). Указанные техники секвенирования обеспечивают определение множества «прочтений» последовательности, которые соответствуют разным участкам одного полинуклеотида. Как правило, известно расстояние между прочтениями или другая информация, касающаяся отношений между прочтениями. В некоторых ситуациях указанные способы секвенирования обеспечивают больше информации по сравнению с секвенированием множества участков последовательности нуклеиновой кислоты случайным образом. При использовании подходящих программных средств для сборки информации последовательности (например, Millikin S.С. et al., Genome Res. 2003, 13: 81-90; Kent, W.J. et al., Genome Res. 2001, 11: 1541-8) возможно применять знания о том, что последовательности не полностью случайны, но, как известно, встречаются на известном расстоянии друг от друга и/или имеют некоторые другие взаимоотношения, и, таким образом, связаны в геноме. Эта информация может помочь в сборке целых последовательностей нуклеиновых кислот в консенсусную последовательность.
2. Нуклеотидные аналоги
Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог применяется в качестве функционального нуклеотидного терминатора (например, в вариантах реализации композиций, способов, наборов и систем, описанных в настоящей заявке). Функциональный нуклеотидный терминатор терминирует полимеризацию нуклеиновой кислоты, например, путем предотвращения участия 3' гидроксильной группы в дополнительных реакциях полимеризации, и содержит функциональную реакционноспособную группу, которая может принимать участие в других химических реакциях с другими химическими фрагментами и группами.
Например, нуклеотидный аналог, содержащий алкинильную группу, применяется согласно некоторым вариантам реализации изобретения, например, нуклеотидный аналог, имеющий структуру в соответствии со следующей структурой:
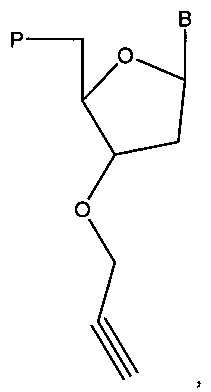
где В представляет собой основание, например, аденин, гуанин, цитозин, тимин или урацил, например, имеющее следующие структуры:
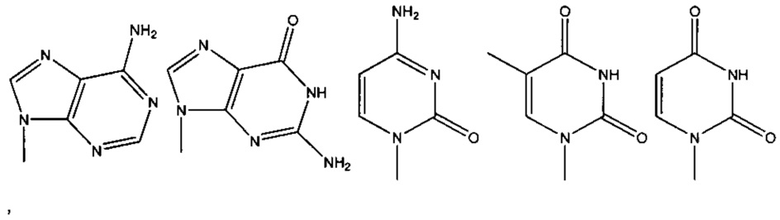
или модифицированное основание или аналог основания, и Р содержит фосфатный фрагмент, например, для получения нуклеотида, имеющего структуру в соответствии со следующей структурой:
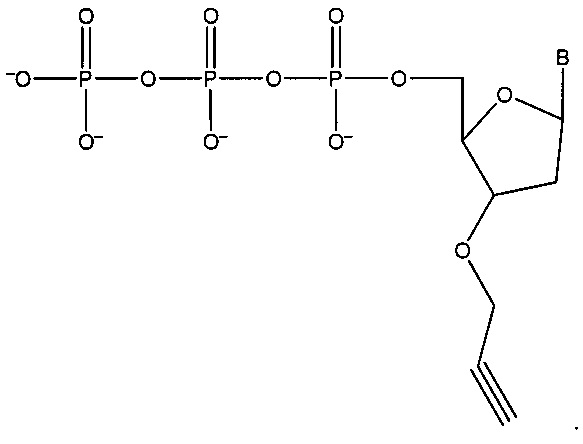
Согласно некоторым вариантам реализации изобретения, Р содержит тетрафосфат; трифосфат; дифосфат; монофосфат; 5' гидроксил; альфа-тиофосфат (например, фосфоротиоат или фосфородитиоат), бетатиофосфат (например, фосфоротиоат или фосфородитиоат), и/или гамма-тиофосфат (например, фосфоротиоат или фосфородитиоат); или альфа-метилфосфонат, бета-метилфосфонат, и/или гамма-метилфосфонат. Согласно некоторым вариантам реализации изобретения, Р содержит азид (например, N3, например, N=N=N), обеспечивая, таким образом, согласно некоторым вариантам реализации изобретения, направленный бифункциональный агент полимеризации. Согласно некоторым вариантам реализации изобретения, технология включает применение нуклеотидного аналога, как описано в одновременно находящейся на рассмотрении заявке н апатент США №14/463,412 и 14/463,416; и международной заявке на патент PCT/US 2014/051726, каждая из которых полностью включена в настоящую заявку посредством ссылки.
Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог представляет собой 3'-О-алкинил-нуклеотидный аналог; согласно некоторым вариантам реализации изобретения, нуклеотидный аналог представляет собой 3'-О-пропаргил-нуклеотидный аналог, такой как 3'-О-пропаргил -dNTP (где N=А, С, G, Τ или U). Пропаргил-нуклеотидный аналог представляет собой нуклеотидный аналог, содержащий основание (например, аденин, гуанин, цитозин, тимин или урацил), дезоксирибозу и алкиновый химический фрагмент, присоединенный к 3'-атому кислорода дезоксирибозы. Химическое связывание между продуктами полимеразного удлинения и подходящими партнерами конъюгации (например, азид-модифицированными молекулами) достигают с высокой эффективностью и специфичностью с использованием например, реакций «клик-химии».
3'-гидроксильная группа нуклеотидного аналога кэпирована химическим фрагментом, например, алкином (например, углерод-углеродной тройной связью), который останавливает дальнейшее удлинение цепи нуклеиновой кислоты (например, ДНК, РНК) при его включении с помощью полимеразы (например, ДНК-или РНК-полимеразы). Алкиновый химический фрагмент представляет собой хорошо известный партнер конъюгации азидной (N3) группы, например, в катализируемой медью (I) реакции 1,3-биполярного циклоприсоединения (например, реакции «клик-химии»). Реакция алкина с азидом приводит к образованию пятичленного триазольного кольца, которое, таким образом, создает ковалентную связь. Связь триазольного кольца при определенной простанственной организации имеет характристики, сходные с характеристиками природной фосфодиэфирной связи, встречающиеся в скелете стандартной нуклеиновой кислоты, и таким образом, триазольная связь имитирует скелет нуклеиновой кислоты. Как предложено в некоторых вариантах реализации в настоящей заявке, применение 3'-О-пропаргил-dNTP приводит к получению фрагментов нуклеиновой кислоты, которые содержат концевую 3'-О-алкиновую группу. Соответственно, указанные фрагменты нуклеиновой кислоты затем можно лигировать химическим путем с использованием реакций «клик-химии» с любыми азид-модифицированными молекулами, такими как 5'-азид-модифицированные олигонуклеотиды (например, такие как адаптеры, предложенные в настоящей заявке, или твердой подложке). Триазольная химическая связь совместима с типичными реакциями и ферментами, используемыми в области биохимии и молекулярной биологии и, соответственно, не ингибирует ферментативные реакции. Соответственно, фрагменты химически лигированной нуклеиновой кислоты можно затем использовать в последующих ферментативных реакциях, таких как полимеразная цепная реакция, реакция секвенирования и т.д.
Согласно некоторым вариантам реализации изобретения, нуклеотидный аналог содержит обратимый терминатор. Например, в нуклеотидной аналоге, содержащем обратимый терминатор, 3' гидроксильные группы кэпированы химическим фрагментом, который может быть удален с помощью специфической химической реакции, с восстановлением, таким образом, свободного 3' гидроксила. Соответственно, некоторые варианты реализации включают реакцию удаления обратимого терминатора и, согласно некоторым вариантам реализации изобретения, дополнительный этап очистки для удаления свободного кэпирующего (терминирующего) фрагмента. Согласно некоторым вариантам реализации изобретения, нуклеотид, содержащий обратимый терминатор, является таким, как описано в заявке на патент США №61/791,730 и/или вмеждународной заявке №PCT/US 14/24391, каждая из которых полностью включена в настоящую заявку посредством ссылки.
3. Адаптеры
Способы технологии включают присоединение адаптера к нуклеиновой кислоте (например, ампликону или «лестнице» фрагментов, как описано в настоящей заявке). Согласно конкретным вариантам реализации изобретения, адаптеры присоединяют к нуклеиновой кислоте с помощью фермента. Фермент может представлять собой лигазу или полимеразу. Лигаза может представлять собой любой фермент, способный лигировать олигонуклеотид (одноцепочечную РНК, двуцепочечную РНК, одноцепочечную ДНК или двуцепочечную ДНК) с другой молекулой нуклеиновой кислоты. Подходящие лигазы включают ДНК-лигазу Т4 и РНК-лигазу Т4 (такие лигазы являются коммерчески доступными, например, из компании New England BioLabs). Способы применения лигаз хорошо известны в данной области техники. Лигирование можно осуществлять по тупым концам или путем использования комплементарных липких концов. Согласно конкретным вариантам реализации изобретения, концы нуклеиновых кислот могут быть фосфорилированными (например, с использованием полинуклеотидкиназы Т4), восстановленным, усеченным (например с помощью экзонуклеазы) или удлиненными (например, с помощью полимеразы и dNTP) для образования тупых концов. При создании тупых концов, указанные концы можно обрабатывать полимеразой и dATP для обеспечения независимого от матрицы присоединения к 3' концам фрагментов, приводящего, таким образом, к получению одного липкого конца А. Этот единственный липкий конец А используется для направления лигирования фрагментов с одним липким концом Τ от 5' конца согласно способу, называемому Т-А клонированием. Полимераза может представлять собой любой фермент, способный добавлять нуклеотиды к 3' и 5' концу молекулы матричной нуклеиновой кислоты.
Согласно некоторым вариантам реализации изобретения, адаптер содержит функциональный фрагмент для химического лигирования с нуклеотидный аналогом. Например, согласно некоторым вариантам реализации изобретения, адаптер содержит азидную группу (например, на 5' конце), которая является реакционноспособной с алкинильной группой (например, пропаргильной группой, например, на 3' конце нуклеиновой кислоты, содержащей нуклеотидный аналог), например, с помощью реакции «клик-химии» (например, с использованием каталитического реагента на основе меди).
Согласно некоторым вариантам реализации изобретения, адаптеры включают универсальную последовательность и/или индекс, например, нуклеотидную последовательность-штрихкод. Дополнительно, адаптеры могут содержать один или более из различных элементов последовательности, включая, но не ограничиваясь указанными, одну или более последовательностей выравнивания праймеров для амплификации или их комплементов, одну или более последовательностей выравнивания праймеров для секвенирования или их комплементов, одну или более штрихкод последовательностей, одну или более последовательностей, общих для множества разных адаптеров или подгрупп разных адаптеров (например, универсальную последовательность), одного или более сайтов распознавания рестрикционным ферментом, одного или более липких концов, комплементарных одной или более мишеням полинуклеотидных липких концов, одного или более сайтов связывания зонда (например для присоединения к платформе для секвенирования, такой как проточная ячейка для массивного параллельного секвенирования, такого как система секвенирования, разработанная компанией Illumina, Inc.), одной или более случайных или почти случайных последовательностей (например, одного или более нуклеотидов, выбранных случайным образом из набора из двух или более разных нуклеотидов в одном или более положениях, где каждый из указанных разных выбранных нуклеотидов находится в одном или более положений, представленных в пуле адаптеров, содержащих случайную последовательность) и их комбинаций. Два или более элементов последовательности могут не являться смежными (например, могут быть разделены одним или более нуклеотидами), являться смежными, частично перекрываются или полностью перекрываться. Например, выравнивающая последовательность праймера для амплификации также может служить в качестве выравнивающей последовательности праймера для секвенирования. Элементы последовательности могут быть расположены на 3' конце или около 3' конца, на 5' конце или около 5' конца или внутри адаптерного олигонуклеотида. Когда адаптерный олигонуклеотид способен образовывать вторичную структуру, такую как структура «шпильки», элементы последовательности могут располагаться частично или полностью за пределами вторичной структуры, частично или полностью внутри вторичной структуры или между последовательностями, принимающими участие в образовании вторичной структуры. Например, когда адаптерный олигонуклеотид содержит структуру «шпильки», элементы последовательности могут быть расположены частично или полностью внутри или снаружи от гибридизуемых последовательностей («стебель»), включая последовательность между гибридизуемыми последовательностями («петля»). Согласно некоторым вариантам реализации изобретения, первые адаптерные олигонуклеотиды из множества первых адаптерных олигонуклеотидов, имеющих разные штрихкод последовательности, включают элемент последовательности, общий для всех первых адаптерных олигонуклеотидов из их множества. Согласно некоторым вариантам реализации изобретения, все вторые адаптерные олигонуклеотиды содержат элемент последовательности, общий для всех вторых адаптерных олигонуклеотидов, то есть отличный от общего с первыми адаптерными олигонуклеотидами элемента последовательности. Различие в элементах последовательности могут представлять собой любые такие различия, при которых по меньшей мере часть разных адаптеров не полностью выравнивается, например, из-за изменений в длине последовательности, делеции или инсерции одного или более нуклеотидов или изменения в нуклеотидном составе в одном или более нуклеотидных положениях (таких как замена основания или модификация основания). Согласно некоторым вариантам реализации изобретения, адаптерный олигонуклеотид содержит 5' липкий конец, 3' липкий конец или 5' липкий конец и 3' липкий конец, комплементарный одному или более полинуклеотидам-мишеням. Комплементарные липкий концы могут иметь длину, составляющую один или более нуклеотидов, включая, но не ограничиваясь указанными 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 или более нуклеотидов. Комплементарные липкие концы могут содержать фиксированную последовательность. Комплементарные липкие концы могут содержать случайную последовательность одного или более нуклеотидов, таких как один или более нуклеотидов, выбранных случайным образом из набора двух или более разных нуклеотидов в одном или более положениях, где каждый из разных выбранных нуклеотидов находится в одном или более положениях, представленных в пуле адаптеров с комплементарными липкими концами, содержащими случайную последовательность. Согласно некоторым вариантам реализации изобретения, липкий конец адаптера комплементарен липкому концу полинуклеотида-мишени, полученного в результате рестрикционного эндонуклеазного переваривания. Согласно некоторым вариантам реализации изобретения, липкий конец адаптера состоит из аденина или тимина.
Согласно некоторым вариантам реализации изобретения, адаптерные последовательности могут содержать элемент идентификации молекулярного сайта связывания для облегчения идентификации и выделения нуклеиновой кислоты-мишени для последующего применения. Молекулярное связывание как механизм аффинности обеспечивает взаимодействие между двумя молекулами с получением стабильного связанного комплекса. Молекулы, которые могут принимать участие в реакциях молекулярного связывания, включают белки, нуклеиновые кислоты, углеводороды, липиды и малые органические молекулы, такие как лиганды, пептиды или лекарственные средства.
Когда молекулярный сайт связывания нуклеиновой кислоты используется как часть адаптера, его можно использовать для селективной гибридизации для выделения последовательности-мишени. Селективная гибридизация может по существу ограничивать гибридизацию с нуклеиновыми кислотами-мишенями, содержащими адаптер с сайтом молекулярного связывания, и захватывающими нуклеиновыми кислотами, которые достаточно комплементарны молекулярному сайту связывания. Таким образом, через «селективную гибридизацию» можно выявить присутствие полинуклеотида-мишени в неочищенном образце, содержащем пул множества нуклеиновых кислот. Пример системы выделения нуклеотид-нуклеотид селективной гибридизации включает систему с несколькими захватывающими нуклеотидами, которые представляют собой комплементарные последовательности к элементам идентификации молекулярного связывания и необязательно иммобилизированы на твердом носителе. Согласно другим вариантам реализации, захватывающие полинуклеотиды могут быть комплементарны последовательностям-мишеням сами по себе или содержать комплементарный штрихкод или уникальную метку в пределах адаптера. Захватывающие полинуклеотиды могут быть иммобилизированы на различных твердых носителях, например, внутри лунки планшета, на гомогенно распределенных сферах, микрочипах или любой другой подходящей поверхности подложки, известной в данной области техники. Гибридизованные комплементарные адаптерные полинуклеотиды, присоединенные к твердому носителю, могут быть выделены путем отмывки нежелательных несвязанных нуклеиновых кислот с сохранением желаемых полинуклеотидов-мишеней. Если комплементарные адаптерные молекулы иммобилизированы на парамагнитных сферах или с помощью других технологий для выделения на основе гранул, то сферы затем можно смешивать в пробирке вместе с полинуклеотидом-мишенью, содержащим адаптеры. После гибридизации адаптерных последовательностей с комплементарными последовательностями, иммобилизированными на сферах, нежелательные молекулы можно вымывать, тогда как сферы сохраняются в пробирке с магнитным или подобным агентом. Желаемые мишени-молекулы затем можно высвобождать путем повышения температуры, изменения рН или с использованием любого другого подходящего способа элюирования, известного в данной области техники.
4. Штрихкоды
Штрихкод представляет собой известную последовательность нуклеиновой кислоты, которая обеспечивает некоторый признак нуклеиновой кислоты, с которой связан штрихкод, подлежащий идентификации. Согласно некоторым вариантам реализации изобретения, признак нуклеиновой кислоты, подлежащий идентификации, представляет собой образец или источник, из которого получена указанная нуклеиновая кислота. Штрихкод-последовательность в целом включает конкретные признаки, которые делают последовательность полезной в реакции секвенирования. Например, штрихкод-последовательности конструируют для получения минимального количества или отсутствия гомополимерных областей, например, 2 или более одинаковых оснований в ряду, таком как АА или ССС, в пределах штрихкод-последовательности. Согласно некоторым вариантам реализации изобретения, штрихкод-последовательности также создают таким образом, что они находятся по меньшей мере на одном редакторском расстоянии от порядка добавления оснований при осуществлении последовательного секвенирования оснований, с гарантией, что первое и последнее основания не совпадают с ожидаемыми основаниями последовательности.
Согласно некоторым вариантам реализации изобретения, штрихкод-последовательности создают таким образом, что каждая последовательность соответствует конкретной нуклеиновой кислоте-мишени, что позволяет снова приводить короткие прочтения последовательности в соответствии с нуклеиновой кислотой-мишенью, из которой они произошли. Способы разработки наборов штрихкод-последовательностей показаны, например, в патенте США №6,235,475, содержания которых полностью включены посредством ссылки в настоящую заявку. Согласно некоторым вариантам реализации изобретения, длина штрихкод-последовательности варьирует от приблизительно 5 нуклеотидов до приблизительно 15 нуклеотидов. Согласно конкретным вариантам реализации, длина штрихкод-последовательности варьирует от приблизительно 4 нуклеотидов до приблизительно 7 нуклеотидов. Поскольку штрихкод-последовательности секвенируют наряду с «лестницей» фрагментов нуклеиновой кислоты, согласно вариантам реализации с использованием более длинных последовательностей, длина штрихкода является минимальной для обеспечения наиболее длинного прочтения с фрагмента нуклеиновой кислоты, присоединенного к штрихкоду. Согласно некоторым вариантам реализации изобретения, штрихкод-последовательности отделены от фрагмента молекулы нуклеиновой кислоты по меньшей мере одним основанием, например, для минимизации гомополимерных комбинаций.
Согласно некоторым вариантам реализации изобретения, длину и последовательность штрихкод-последовательностей конструируют для достижения желаемого уровня точности определения идентичности нуклеиновой кислоты. Например, согласно некоторым вариантам реализации изобретения, штрихкод-последовательности конструируют таким образом, что после введения приемлемого количества точечных мутаций, идентичность связанной нуклеиновой кислоты все еще может быть установлена с желаемой точностью. Согласно некоторым вариантам реализации изобретения, Τn-5 трнаспозаза (коммерчески доступная из компании Epicentre Вiотехнологии; Мэдисон, Висконсин) разрезает нуклеиновую кислоту на фрагменты и встраивает короткие кусочки ДНК в область разрезов. Короткие кусочки ДНК используют для встраивания штрихкод-последовательностей.
Способ присоединения адаптеров, содержащий штрихкоды, к матрицам нуклеиновой кислоты, описан в публикации заявки на патент США №2008/0081330 и международной патентной заявке №PCT/US 09/64001, содержание каждой из которых полностью включено посредством ссылки в настоящую заявку. Способы разработки наборов штрихкод последовательностей и другие способы присоединения адаптеров (например, содержащих штрихкод-последовательности) показаны в патентах США №6,138,077; 6,352,828; 5,636,400; 6,172,214; 6235,475; 7,393,665; 7,544,473; 5,846,719; 5,695,934; 5,604,097; 6,150,516; RE39,793; 7,537,897; 6172,218; и 5,863,722, содержание каждого из которых полностью включено посредством ссылки в настоящую заявку. Согласно конкретным вариантам реализации изобретения, один штрихкод присоединен к каждому фрагменту. Согласно другим вариантам реализации, множество штрихкодов, например, два штрихкода, присоединено к каждому фрагменту.
5. Образцы
Согласно некоторым вариантам реализации изобретения, молекулы матричной нуклеиновой кислоты (например, ДНК или РНК) выделены из биологического образца, содержащего различные другие компоненты, такие как белки, липиды и нематричные нуклеиновые кислоты. Молекулы матричной нуклеиновой кислоты могут быть получены из любого материала (например, клеточного материала (живого или неживого), внеклеточного материала, вирусного материала, образцов окружающей среды (например, метагеномных образцов), синтетического материала (например, ампликонов, таких как ампликоны, полученные с помощью ПЦР или других технологий амплификации)), полученного из животного, растения, бактерии, архей, грибов или любого другого организма. Биологические образцы для применения согласно настоящему изобретению включают вирусные частицы или их препараты. Молекулы матричной нуклеиновой кислоты могут быть получены непосредственно из организма или из биологического образца, полученного из организма, например, из крови, мочи, спинномозговой жидкости, семенной жидкости, слюны, мокроты, стула, волос, пота, слезной жидкости, кожи и ткани. Типичные образцы включают, но не ограничиваются указанными, цельную кровь, лимфатическую жидкость, сыворотку, плазму, буккальные клетки, пот, слезную жидкость, слюну, мокроту, волосы, кожу, биопсию, спинномозговую жидкость (СМЖ), амниотическую жидкость, семенную жидкость, вагинальные выделения, серозную жидкость, синовиальную жидкость, перикардиальную жидкость, перитонеальную жидкость, плевральную жидкость, транссудаты, экссудаты, кистозную жидкость, желчь, мочу, желудочные жидкости, кишечные жидкости, образцы фекалий и мазки, пунктаты (например, костномозговые, тонкоигольные и т.д.), смывы (например, оральный, носоглоточный, бронхиальный, бронхоальвеолярный смыв, глазной, ректальный, кишечный, вагинальный, эпидермальный и т.д.) и/или другие образцы.
Любой образец ткани или жидкости организма может использоваться в качестве источника нуклеиновой кислоты для применения в технологии, включая судебные образцы, архивированные образцы, фиксированные образцы и/или образцы, хранимые в течение длительных периодов времени, например, свежезамороженные, фиксированные метанолом/уксусной кислотой или зафиксированные формалином и залитые парафином (FFPE) образцы и пробы. Молекулы матрицы нуклеиновой кислоты также могут быть выделены из культивируемых клеток, таких как клетки первичной культуры или клеточной линии. Клетки или ткани, из которых получают матричные нуклеиновые кислоты, могут быть инфицированы вирусом или другим внутриклеточным патогеном. Образец также может представлять собой тотальную РНК, экстрагированную из биологического образца, библиотеки кДНК, вирусной или геномной ДНК. Образец также может представлять собой выделенную ДНК неклеточной природы, например амплифицированную/изолированную ДНК, которую хранили в холодильнике.
Молекулы матрицы нуклеиновой кислоты могут быть получены, например, путем экстракции из биологического образца, например, с помощью различных способов, таких как способы, описанные в источнике Maniatis, et al. (1982) Molecular Cloning: A Laboratory Manual, Cold Spring Harbor, N.Y. (см., например, с. 280-281).
Согласно некоторым вариантам реализации изобретения, осуществляют выбор размера нуклеиновых кислот с удалением очень коротких фрагментов или очень длинных фрагментов. Подходящие способы выбора размера известны в данной области техники. Согласно некоторым вариантам реализации, размер ограничен 0,5, 1, 2, 3, 4, 5, 7, 10, 12, 15, 20, 25, 30, 50, 100 т.п.н. или более длинных.
Согласно некоторым вариантам реализации, нуклеиновую кислоту амплифицируют. Могут использоваться любые способы амплификации, известные в данной области техники. Примеры способов амплификации, которые можно использовать, включают, но не ограничиваются указанными, ПЦР, количественную ПЦР, количественную флуоресцентную ПЦР (QF-PCR), множественную флуоресцентную ПЦР (MF-PCR), ПЦР в реальном времени (RT-PCR), одноклеточную ПЦР, ПЦР длины полиморфизма рестрикционного фрагмента (PCR-RFLP), ПЦР с горячим стартом, «вложенной» ПЦР, in situ ПЦР молекулярных колоний, in situ амплификацию по типу катящегося колеса (RCA), мостиковую ПЦР, пикотитрационную ПЦР и эмульсионную ПЦР. Другие подходящие способы амплификации включают лигазную цепную реакцию (LCR), транскрипционную амплификацию, самоподдерживающуюся репликацию последовательности, селективную амплификацию полинуклеотидных последовательностей-мишеней, праймированную консенсусной последовательностью полимеразную цепную реакцию (CP-PCR), произвольно праймированную полимеразную цепную реакцию (AP-PCR), праймированную вырожденным олигонуклеотидом ПЦР (DOP-PCR) и амплификацирующее секвенирвоание на основе нуклеиновой кислоты (NABSA). Другие способы амплификации, которые могут использоваться в настоящей заявке, включают способы, описанные в патентах США №5,242,794; 5,494,810; 4,988,617 и 6,582,938.
Согласно некоторым вариантам реализации изобретения, восстановление концов осуществляют для создания тупого конца 5' фосфорилированной нуклеиновой кислоты с использованием коммерческих наборов, таких как наборы, доступные из компании Epicentre Bioтехнологии (Мэдисон, Висконсин).
6. Секвенирование нуклеиновой кислоты
Согласно некоторым вариантам реализации технологии, получают данные последовательности нуклеиновой кислоты. Различные варианты реализации платформ для секвенирования нуклеиновой кислоты (например, секвенатор нуклеиновой кислоты) включают компоненты, описанные ниже. Согласно различным вариантам реализации, инструмент секвенирования включает жидкостную доставку и контрольную единицу, единицу обработки образца, единицу выявления сигнал и единицу получения данных и анализа и контрольную единицу. Различные варианты реализации инструмента обеспечивают автоматизированное секвенирование, которое используется для сбора информации последовательности из множества последовательностей параллельно и/или по существу одновременно.
Согласно некоторым вариантам реализации изобретения, жидкостная доставка и контрольная единица включает систему доставки реагента. Система доставки реагента включает резервуар реагента для хранения различных реагентов. Реагенты могут включать праймеры на основе РНК, прямые/обратные ДНК праймеры, нуклеотидные смеси (например, композиции, содержащие нуклеотидные аналоги, как предложено в настоящей заявке) для секвенирования путем синтеза, буферы, промывочные реагенты, блокирующие реагенты, нейтрализующие заряд реагенты и т.п. Дополнительно система доставки реагента может включать систему пипетирования или систему непрерывного ввода, которая связывает единицу обработки образца с резервуаром реагента.
Согласно некоторым вариантам реализации изобретения, единица обработки образца включает камеру для образца, такую как проточная ячейка, субстрат, микрочип, многолуночный планшет и т.д. Единица обработки образца может включать множество дорожек, множество каналов, множество лунок или других вариантов для по существу одновременной обработки наборов множества образцов. Кроме того, единица обработки образца может включать множество камер для образцов для обеспечения обработки множества циклов одновременно. Согласно конкретным вариантам реализации изобретения, система может осуществлять выявление сигнала в одной камере для образце при фактически одновременной обработке другой камеры для образца. Кроме того, единица обработки образца может включать автоматическую систему для перемещения или произведения манипуляций с камерой образца. Согласно некоторым вариантам реализации изобретения, единица выявления сигнала может включать сенсор изображения или выявления. Например, сенсор изображения или выявления (например, детектор флуоресценции или электрический детектор) может включать CCD, CMOS, ионный сенсор, такой как ионный чувствительный слой, наносимый на CMOS, гальванометр и т.д. Единица выявления сигнала может включать систему возбуждения для стимуляции испускания сигнала меткой, такой как флуоресцентный краситель. Система выявления может включать источник излучения, такой как дуговая лампа, лазер, светодиод (LED) и т.д. Согласно конкретным вариантам реализации изобретения, единица выявления сигнала включает оптику для пропускания света от источника излучения к образцу или от образца к сенсору изображения или выявления. Альтернативно, единица выявления сигнала может не включать источник излучения, например, когда сигнал производится спонтанно в результате реакции секвенирования. Например, сигнал может появляться в результате взаимодействия высвобождаемого фрагмента, такого как высвобождаемый ион, взаимодействующий с ион-чувствительный слоем, или пирофосфат, реагирующий с ферментом или другим катализатором с получением хемилюминесцентного сигнала. В другом примере изменения электрического тока, напряжения или сопротивления выявляют без необходимости источника излучения.
Согласно некоторым вариантам реализации изобретения, единица анализа получения данных и единица контроля отслеживают различные параметры системы. Параметры системы могут включать температуру различных частей инструмента, таких как единица обработки образца или резервуары для реагента, объемы различных реагентов, статус различных субкомпонентов системы, таких как манипулятор, шаговый двигатель, насос и т.д или любую их комбинацию.
Специалистам в данной области техники очевидно, что различные варианты реализации инструментов и систем используются для осуществления на практике способов секвенирования, таких как секвенирования путем синтеза, одномолекулярные способы секвенирования и другие способы секвенирования. Секвенирование путем синтеза может включать встраивание меченных краской нуклеотидов, обрыв цепи, ионное/протонное секвенирование, пирофосфатное секвенирование и т.д. Одномолекулярные способы могут включать ступенчатое секвенирования, где реакции секвенирования приостанавливаются для определения идентичности включенного нуклеотида.
Согласно некоторым вариантам реализации изобретения, инструмент секвенирования определяет последовательность нуклеиновой кислоты, такую как полинуклеотид или олигонуклеотид. Нуклеиновая кислота может включать ДНК или РНК и может быть одноцепочечной, такой как оцДНК и РНК или двуцепочечной, такой как дцДНК или пара РНК/кДНК. Согласно некоторым вариантам реализации изобретения, нуклеиновая кислота может включать или происходить из библиотеки фрагментов, библиотеки партнеров, фрагмента СhIР и т.д. Согласно конкретным вариантам реализации изобретения, инструмент секвенирования может получать информацию последовательности от одной молекулы нуклеиновой кислоты или от группы по существу идентичных молекул нуклеиновой кислоты.
Согласно некоторым вариантам реализации изобретения, инструмент секвенирования может выводить данные прочтения последовательности нуклеиновой кислоты в виде файлов различных типов/форматов разных выходных данных, включая, но не ограничиваясь указанными: *.txt, *.fasta, *.csfasta, *seq.txt, *qseq.txt, *.fastq, *.sff, *prb.txt, *.sms, *srs, и/или *.qv.
7. Технологии секвенирования нового поколения
Конкретные технологии секвенирования, рассматриваемые в соответствии с технологией, представляют собой способы секвенирования нового поколения (NGS), которые обладают общими признаками массового параллелизма, направлены на высокую производительность с целью обеспечения более низкой стоимости по сравнению с предыдущими способами секвенирования (см., например, Voelkerding et al., Clinical Chem., 55: 641-658, 2009; MacLean et al., Nature Rev. Microbiol., 7: 287-296; каждый из которых полностью включен в настоящую заявку посредством ссылки). NGS способы можно в целом разделить на способы, в которых, как правило, используется матрица для амплификации, и способы, в которых не используется матрица для амплификации. Способы, для которых требуется амплификация, включают технологию пиросеквенирования, коммерчески доступную от компании Roche в виде платформы для технологии 454 (например, GS 20 и GS FLX), в виде платформы Solexa, коммерчески доступной от компании Illumina, и платформы Supported Oligonucleotide Ligation and Detection (SOLiD), коммерчески доступной от компании Applied Biosystems. Примером не связанных с амплификацией способов, также известных как одномолекулярное секвенирование, является платформа HeliScope, коммерчески доступная от компании Helicos BioSciences, и появляющиеся платформы, коммерчески доступные от компании VisiGen, Oxford Nanopore Technologies Ltd., Life Technologies/Ion Torrent и Pacific Biosciences соответственно.
При пиросеквенировании (Voelkerding et al., Clinical Chem., 55: 641-658, 2009; MacLean et al., Nature Rev. Microbiol., 7: 287-296; патент США №6,210,891; патент США №6,258,568; каждый из которых полностью включен в настоящую заявку посредством ссылки) библиотеки фрагментов NGS клонально амплифицируют in-situ путем захвата одной молекулы матрицы с помощью гранул, покрытых олигонуклеотидами, комплементарными адаптерам. Каждая гранула, содержащая матрицу одного типа, помещается в микропузырек по типу «вода в масле» и матрицу клонально амплифицируют с использованием способа, называемого эмульсионной ПЦР. После амплификации эмульсию разрушают и гранулы складывают в отдельные лунки титрационного пикопланшета, действующего в качестве проточной ячейка во время реакций секвенирования. Упорядоченное многократное введение каждого из четырех реагентов dNTP в проточную ячейку происходит в присутствии ферментов секвенирования и люминесцентного репортера, такого как люцифераза. В случае, когда подходящий dNTP добавляют к 3' концу праймера секвенирования, продуцирующийся в результате АТР вызывает вспышку люминесценции в пределах лунки, которая регистрируется с использованием камеры CCD. Возможно достигать длину прочтений, составляющую более чем или равную 400 основаниям, и можно достичь получения 106 прочтений последовательности, что приводит к получению до 500 миллионов пар оснований (мегабаз) последовательности.
На платформе Solexa/Illumina (Voelkerding et al., Clinical Chem., 55: 641-658, 2009; MacLean et al., Nature Rev. Microbiol., 7: 287-296; патент США №6,833,246; патент США №7,115,400; патент США №6,969,488; каждый из которых полностью включен в настоящую заявку посредством ссылки) данные секвенирования производятся в виде коротких прочтений. В указанном способе фрагменты библиотеки фрагментов NGS захватываются на поверхность проточной ячейки, которая покрыта олигонуклеотидными якорными молекулами. Якорным молекулы используется в качестве ПЦР-праймера, но из-за длины матрицы и его близости к другим близлежащим якорным олигонуклеотидам, удлинение с помощью ПЦР приводит к образованию «свода» молекулы с ее гибридизацией с соседним якорным олигонуклеотидом и образованием мостиковой структуры на поверхности проточной ячейки. Указанные петли ДНК денатурируют и расщепляют. Прямые цепи затем секвенируют с помощью обратимо окрашенных терминаторов. Включенные в последовательность нуклеотиды определяют по выявлению флуоресценции после включения, где каждый флуоресцентный и блокирующий агент удаляется до следующего цикла добавления dNTP. Длина прочтения последовательности варьирует от 36 нуклеотидов до более чем 100 нуклеотидов с общим выходом, превышающим 1 биллион пар нуклеотидов на рабочий цикл анализа.
Секвенирование молекул нуклеиновой кислоты с использованием технологии SOLiD (Voelkerding et al., Clinical Chem., 55: 641-658, 2009; MacLean et al., Nature Rev. Microbiol., 7: 287-296; патент США №5,912,148; патент США №6,130,073; каждый из которых полностью включен в настоящую заявку посредством ссылки) также включает клональную амплификацию библиотеки фрагментов NGS с помощью эмульсионной ПЦР. После этого гранулы, содержащие матрицу, иммобилизируют на дериватизированной поверхности стеклянной проточной ячейки и отжигают с праймером, комплементарным адаптерному олигонуклеотиду. Однако вместо использования указанного праймера для 3' удлинения он используется для получения 5' фосфатной группы для лигирования для проверочными зондами, содержащими два зонд-специфичных основания с последующими 6 вырожденными основаниями и одной из четырех флуоресцентных меток. В системе SOLiD проверочные зонды имеют 16 возможных комбинаций двух оснований на 3' конце каждого зонда и одного из четырех флуоресцентных красителей на 5' конце. Цвет флуоресцентного красителя и, таким образом, идентичность каждого зонда, соответствует определенной схеме кодирования цветового пространства. После множества циклов (как правило, 7) выравнивания зонда, лигирования зонда и выявления флуоресцентного сигнала следует денатурация и затем второй цикл секвенирования с использованием праймера, который смещен на одно основание по сравнению с исходным праймером. Указанным образом последовательность матрицы можно реконструировать путем вычисления; основания матрицы проверяются дважды, что приводит к повышению точности. Длина прочтения последовательности составляет в среднем 35 нуклеотидов, и общий выход превышает 4 биллиона оснований на рабочий цикл секвенирования.
Согласно конкретным вариантам реализации изобретения, используют HeliScope от компании Helicos BioSciences (Voelkerding et al., Clinical Chem., 55: 641-658, 2009; MacLean et al., Nature Rev. Microbiol., 7: 287-296; патент США №7,169,560; патент США №7,282,337; патент США №7,482,120; патент США №7,501,245; патент США №6,818,395; патент США №6,911,345; патент США №7,501,245; каждый из которых полностью включен в настоящую заявку посредством ссылки). Секвенирование достигают путем добавления полимеразы и серийных добавлений флуоресцентно-меченных dNTP реагентов. Включение приводят к появлению флуоресцентного сигнала, соответствующего dNTP, и указанный сигнал захватывается камерой CCD до каждого цикла добавления dNTP. Длина прочтения последовательности варьирует от 25-50 нуклеотидов с общим выходом, превышающим 1 биллион пар нуклеотидов на аналитический рабочий цикл.
Согласно некоторым вариантам реализации изобретения, используют систему секвенирования 454 от компании Roche (Margulies et al. (2005) Nature 437: 376-380). Секвенирования 454 включает два этапа. В первом этапе ДНК разрезают на фрагменты, составляющие приблизительно 300-800 пар оснований, и указанные фрагменты имеют тупые концы. Олигонуклеотидные адаптеры затем лигируют с концами фрагментов. Адаптер служат в качестве праймеров для амплификации и секвенирования фрагментов. Фрагменты могут быть присоединены к ДНК-захватывающим гранулам, например, покрытым стрептавиином гранулам, с использованием, например, адаптера, который содержит 5'-биотиновую метку. Фрагменты, присоединенные к гранулам, амплифицируют с помощью ПЦР в пределах капель масляно-водной эмульсии. Результатом является множество копий клонально амплифицированный фрагментов ДНК на каждой грануле. На втором этапе гранулы захватывают в лунки (объемом в несколько пиколитров). Пиросеквенирование осуществляют на каждом ДНК-фрагменте параллельно. Добавление одного или более нуклеотидов приводит к генерации светового сигнала, который регистрируется на камеру CCD инструмента секвенирования. Интенсивность сигнала пропорциональна количеству включенных нуклеотидов. Пиросеквенирование используют пирофосфат (PPi), который высвобождается при добавлении нуклеотида. PPi превращается в АТР с помощью АТР-сульфурилазы в присутствии аденозин 5' фосфосульфата. Люцифераза использует АТР для превращения люциферина в оксилюциферин, и в результате указанной реакции генерируется свет, который выявляется и анализируется.
Технология Ion Torrent представляет собой способ ДНК-секвенирования на основе выявления ионов водорода, которые высвобождаются во время полимеризации ДНК (см., например, Science 327(5970): 1190 (2010); публикации заявок на патент США №20090026082, 20090127589, 20100301398, 20100197507, 20100188073, и 20100137143, полностью включенные посредством ссылки во всех отношениях). Микролунка содержит фрагмент библиотеки фрагментов NGS, подлежащей секвенированию. Под слоем микролунок находится гиперчувствительный ионный сенсор ISFET. Все слои содержатся в пределах полупроводящего чипа CMOS, подобного чипу, используемому в электронной промышленности. Когда dNTP включается в растущую комплементарную цепь, высвобождается ион водорода, который возбуждает гиперчувствительный ионный сенсор. Если гомополимерные повторы присутствуют в последовательности матрицы, множество dNTP молекул будут включены в один цикл. Это приводит к соответствующему количеству высвобождаемых атомов водорода и пропорционально более высокому электрическому сигналу. Указанная технология отличается от других технологий секвенирования, в которых не используются модифицированные нуклеотиды или оптические устройства. Точность на основание секвенатора Ion Torrent составляет ~99,6% для прочтений из 50 оснований, при этом за рабочий цикл генерируется ~100 мегабаз. Длина прочтения составляет 100 пар оснований. Точность гомополимерных повторов размером 5 повторов в длину составляет ~98%. Преимуществами ионного полупроводникового секвенирования является быстрая скорость секвенирования и малая предоплата и производственные затраты. Однако стоимость получения устройства рН-опосредованного секвенирования составляет приблизительно $50000, за исключением оборудования, необходимого для получения образца, и сервера для данного анализа.
Другой типичный подход к секвенированию нуклеиновой кислоты, который может быть адаптирован для применения в соответствии с настоящим изобретением, был разработан компанией Stratos Genomics, Inc. и включает применение X-пандомеров. Указанный процесс секвенирования, как правило, включает обеспечения дочерней цепи, полученной в результате синтеза, направляемого матрицей. Дочерняя цепь в целом включает множество субъединиц, соединенных в последовательность, соответствующую непрерывной нуклеотидной последовательности всей или части нуклеиновой кислоты-мишени, в которой отдельные субъединицы включают связывающий домен, по меньшей мере одну пробу или остаток нуклеинового основания и по меньшей мере одну селективно расщепляемую связь. Селективно расщепляемая связь (связи) расщепляется (расщепляются) с получением Х-пандомера, более длинного, чем множество субъединиц дочерней цепи. Х-пандомер, как правило, включает созывающий домен и репортерные элементы для анализа генетической информации в последовательности, соответствующей непрерывной нуклеотидной последовательности всей или части нуклеиновой кислоты-мишени. Репортерные элементы Х-пандомера затем выявляют. Дополнительные подробности основанного на Х-пандомерах подхода описаны, например, в публикации патента США №20090035777 под названием «HIGH THROUGHPUT NUCLEIC ACID SEQUENCING BY EXPANSION,» поданного 19 июня 2008 г., который полностью включен в настоящую заявку.
Другие способы одномолекулярного секвенирования включают секвенирование в реальном времени путем синтеза с использованием платформы VisiGen (Voelkerding et al., Clinical Chem., 55: 641-58, 2009; патент США №7,329,492; заявка на патент США №11/671956; заявка на патент США №11/781166; каждый из которых полностью включен в настоящую заявку посредством ссылки), согласно которому фрагменты библиотеки NGS иммобилизируют, праймируют и затем подвергают удлинению цепи с использованием флуоресцентно-модифицированной полимеразы и флуоресцентной акцепторной молекулы, что приводит к выявляемому резонансному переносу энергии флуоресценции (FRET) при добавлении нуклеотида.
Другая система одномолекулярного секвенирования в реальном времени, разработанная компанией Pacific Biosciences (Voelkerding et al., Clinical Chem., 55: 641-658, 2009; MacLean et al., Nature Rev. Microbiol., 7: 287-296; патент США №7,170,050; патент США №7,302,146; патент США №7,313,308; патент США №7,476,503; все из которых включены в настоящую заявку посредством ссылки), использует реакционные лунки диаметром 50-100 нм и включает реакционный объем, составляющий приблизительно 20 зептолитров (10-21 л). Реакции секвенирования проводят с использованием иммобилизированной матрицы, модифицированной ДНК-полимеразой phi29, и высокой локальной концентрации флуоресцентно меченных dNTP. Высокая локальная концентрация и постоянные условия реакции позволяют зафиксировать события включения в реальном времени путем выявления флуоресцентного сигнала с использованием лазерного возбуждения, светового волновода и камеры CCD.
Согласно конкретным вариантам реализации изобретения, используют способы одномолекулярного секвенирования ДНК в реальном времени (SMRT) с использованием волноводов с нулевой модой (ZMW), разработанных компанией Pacific Biosciences, или подобных способов. При использовании указанной технологии ДНК секвенируют на чипах SMRT, каждый из которых содержит тысячи волноводов с нулевой модой (ZMW). ZMW представляет собой дыру диаметром в десятки нанометров, сделанную в 100 нм металлической пленке, помещенной на субстрат на основе диоксила кремния. Каждый ZMW становится нанофотонной камерой визуализации, обеспечивающей зону выявления, составляющую всего 20 зептолитров (10-21 л). При указанной зоне активность одной молекулы можно выявить среди фона тысяч меченных нуклеотидов. ZMW обеспечивает окно для наблюдения осуществления ДНК-полимеразой секвенирования путем синтеза. В пределах каждой камеры одна молекула ДНК-полимеразы присоединяется к поверхности дна таким образом, что она все время сохраняется в пределах зоны выявления. Фосфосвязанные нуклеотиды, каждый тип которых помечен разными окрашенными флуорофорами, затем вводят в реакционный раствор в высокой концентрации, которая обеспечивает скорость, точность и производительность. Из-за малого размера ZMW даже при указанных высоких биологически приемлемых концентрациях зона выявления занята нуклеотидами только малую часть времени. Кроме того, пребывание в зоне выявления недолгое и длится только несколько микросекунд из-за очень малого расстояния, которое нуклеотиды должны преодолеть для перемещения путем диффузии. Результатом является очень низкий фон.
Согласно некоторым вариантам реализации изобретения, используют нанопоровое секвенирование (Soni GV и Meller Α. (2007) Clin Chem 53: 1996-2001). Нанопора представляет собой небольшую дыру диаметром порядка 1 нанометра. Погружение нанопоры в проводящую жидкость и приложение к ней потенциала приводит к слабому электрическому току из-за проведения ионов через нанопору. Количество проходящего тока зависит от размера нанопоры. Так как молекула ДНК проходит через нанопору, каждый нуклеотид на молекуле ДНК загораживает нанопору в разной степени. Таким образом, изменение тока, проходящего через нанопору по мере прохождения молекулы ДНК через нанопору, обеспечивает считывание последовательности ДНК.
Согласно некоторым вариантам реализации изобретения, способ секвенирования использует химически-чувствительный полевой транзисторный (chemFET) чип для секвенирования ДНК (например, как описано в публикации заявки на патент США №20090026082). В одном примере способа молекулы ДНК помещают в реакционные камеры и молекулы матрицы гибридизуют с праймером для секвенирования, связанным с полимеразой. Включение одного или более трифосфатов в новую цепь нуклеиновой кислоты на 3' конце праймера для секвенирования можно выявить путем изменения тока с помощью chemFET. Чип может иметь множество сенсоров chemFET. В другом примере отдельные нуклеиновые кислоты могут быть присоединены к гранулам и амплифицированы на гранулах, и отдельные гранулы можно перемещать в отдельные реакционные камеры на чипе chemFET, где каждая указанная камера имеет сенсор chemFET, и нуклеиновые кислоты могут быть секвенированы.
Согласно некоторым вариантам реализации изобретения, в технике секвенирования используется электронный микроскоп (Moudrianakis Ε.N. и Beer M. Proc Natl Acad Sci USA. 1965 March; 53: 564-71). В одном примере техники отдельные молекулы ДНК являются меченными с использованием метаболических меток, которые можно отличить с помощью пользованием электронного микроскопа. Указанные молекулы затем растягиваются на плоской поверхности и визуализируются с использованием электронного микроскопа для измерения последовательностей.
Согласно некоторым вариантам реализации изобретения, используется «четырехцветное секвенирование путем синтеза с использованием расщепляемого флуоресцентных нуклеотидных обратимых терминаторов», как описано в источнике Turro, et al. PNAS 103: 19635-40 (2006), например, система, коммерчески доступная из компании Intelligent Bio-Systems. Технология, описанная в публикациях заявок на патент США №2010/0323350, 2010/0063743, 2010/0159531, 20100035253, 20100152050, включена в настоящую заявку посредством ссылки во всех отношениях.
Процессы и системы для такого секвенирования в реальном времени, которые могут быть адаптирована для применения в соответствии с изобретением, описаны, например, в патенте США №. 7,405,281, под названием «Fluorescent nucleotide analogs and uses therefor», опубликованном 29 июля 2008 г., Xu et al.; 7,315,019, под названием «Arrays of optical confinements and uses thereof», опубликованном 1 января 2008 г., Turner et al.; 7,313,308, под названием «Optical analysis of molecules», опубликованном 25 декабря 2007 г. Turner et al.; 7,302,146, под названием «Apparatus and method for analysis of molecules», опубликованном 27 ноября 2007 г. Turner et al.; и 7,170,050, под названием «Apparatus and methods for optical analysis of molecules», опубликованном 30 января 2007 г. Turner et al.; и публикациях патентов США №20080212960, под названием «Methods and systems for simultaneous real-time monitoring of optical signals from multiple sources», поданного 26 октября 2007 г. Lundquist et al.; 20080206764, под названием «Flowcell system for single molecule detection», поданного 26 октября 2007 г. Williams et al.; 20080199932, под названием «Active surface coupled polymerases)), поданного 26 октября 2007 г. Hanzel et al.; 20080199874, под названием ((CONTROLLABLE STRAND SCISSION OF MINI CIRCLE DNA», поданного 11 февраля 2008 г. Otto et al.; 20080176769, под названием «Articles having localized molecules disposed thereon and methods of producing same», поданного 26 октября 2007 г. Rank et al.; 20080176316, под названием «Mitigation of photodamage in analytical reactions», поданного 31 октября 2007 г. Eid et al.; 20080176241, под названием «M Mitigation of photodamage in analytical reactions», поданного 31 октября 2007 г. Eid et al.; 20080165346, под названием «Methods and systems for simultaneous real-time monitoring of optical signals from multiple sources», поданного 26 октября 2007 г. Lundquist et al.; 20080160531, под названием «Uniform surfaces for hybrid material substrates and methods for making and using same», поданного 31 октября 2007 Korlach; 20080157005, под названием «Methods and systems for simultaneous real-time monitoring of optical signals from multiple sources», поданного 26 октября 2007 г. Lundquist et al.; 20080153100, под названием «Articles having localized molecules disposed thereon and methods of producing same», поданного 31 октября 2007 Rank et al.; 20080153095, под названием «CHARGE SWITCH NUCLEOTIDES)), поданного 31 октября 2007 Williams et al.; 20080152281, под названием «Substrates, systems and methods for analyzing materials)), поданного 31 октября 2007 Lundquist et al.; 20080152280, под названием «Substrates, systems and methods for analyzing materials)), поданного 31 октября 2007 Lundquist et al.; 20080145278, под названием «Uniform surfaces for hybrid material substrates and methods for making and using same», поданного 31 октября 2007 Korlach; 20080128627, под названием «SUBSTRATES, SYSTEMS AND METHODS FOR ANALYZING MATERIALS», поданного 31 августа 2007 г. Lundquist et al.; 20080108082, под названием ((Polymerase enzymes and reagents for enhanced nucleic acid sequencing)), поданного 22 октября 2007 г. Rank et al.; 20080095488, под названием «SUBSTRATES FOR PERFORMING ANALYTICAL REACTIONS», поданного 11 июня 2007 г. Foquet et al.; 20080080059, под названием «MODULAR OPTICAL COMPONENTS AND SYSTEMS INCORPORATING SAME», поданного 27 сентября 2007 г. Dixon et al.; 20080050747, под названием «Articles having localized molecules disposed thereon and methods of producing and using same», поданного 14 августа 2007 г. by Korlach et al.; 20080032301, под названием «Articles having localized molecules disposed thereon and methods of producing same», поданного 29 марта 2007 г. Rank et al.; 20080030628, под названием «Methods and systems for simultaneous real-time monitoring of optical signals from multiple sources», поданного 9 февраля 2007 г. Lundquist et al.; 20080009007, под названием «CONTROLLED INITIATION OF PRIMER EXTENSION», поданного 15 июля 2007 г. Lyle et al.; 20070238679, под названием «Articles having localized molecules disposed thereon and methods of producing same», поданного 30 марта 2006 г. Rank et al.; 20070231804, под названием «Methods, systems and compositions for monitoring enzyme activity and applications thereof», поданного 31 марта 2006 г. Korlach et al.; 20070206187, под названием «Methods and systems for simultaneous real-time monitoring of optical signals from multiple sources», поданного 9 февраля 2007 г. Lundquist et al.; 20070196846, под названием «Polymerases for nucleotide analog incorporation», поданного 21 декабря 2006 г. Hanzel et al.; 20070188750, под названием «Methods and systems for simultaneous real-time monitoring of optical signals from multiple sources», поданной 7 июля 2006 г. Lundquist et al.; 20070161017, под названием «MITIGATION OF PHOTODAMAGE IN ANALYTICAL REACTIONS», поданного 1 декабря 2006 г. Eid et al.; 20070141598, под названием «Nucleotide Compositions and Uses Thereof», поданного 3 ноября 2006 г. Turner et al.; 20070134128, под названием «Uniform surfaces for hybrid material substrate and methods for making and using same», поданного 27 ноября 2006 г. Korlach; 20070128133, под названием «Mitigation of photodamage in analytical reactions», поданного 2 декабря 2005 г. Eid et al.; 20070077564, под названием «Reactive surfaces, substrates and methods of producing same», поданного 30 сентября 2005 г. Roitman et al.; 20070072196, под названием «Fluorescent nucleotide analogs and uses therefore», поданного 29 сентября 2005 г. Xu et al; и 20070036511, под названием «Methods and systems for monitoring multiple optical signals from a single source», поданной 11 августа 2005 г. Lundquist et al.; и Korlach et al. (2008) «Selective aluminum passivation for targeted immobilization of single DNA polymerase molecules in zero-mode waveguide nanostructures» PNAS 105(4): 1176-81, все из который полностью включены в настоящую заявку посредством ссылки.
8. Анализ последовательности нуклеиновой кислоты
Согласно некоторым вариантам реализации изобретения, компьютерная аналитическая программа используется для перевода сырых данных, полученных в результате детектирующего анализа (например, секвенирования прочтений), в данные прогностической ценности для конечного пользователя (например, медицинского персонала). Пользователь может иметь доступ к прогностическим данным с помощью любых подходящих способов. Таким образом, согласно некоторым предпочтительным вариантам реализации, настоящая технология обеспечивает дополнительное преимущество, которое заключается в том, что пользователь, который, вероятно, не имеет опыта в области генетики или молекулярной биологии, не должен понимать сырые данные. Данные предоставляются непосредственно конечному пользователю в наиболее полезно виде. Пользователь затем может сразу использовать информацию для определения полезной информации (например, в медицинской диагностике, исследовании или скрининге).
Согласно некоторым вариантам реализации изобретения, предложена система для реконструкции последовательности нуклеиновой кислоты. Система может включать секвенатор нуклеиновых кислот, накопитель данных образца последовательности, накопитель данных референсной последовательности и аналитическое вычислительное устройство/сервер/узел. Согласно некоторым вариантам реализации изобретения, аналитическое вычислительное устройство/сервер/узел может представлять собой рабочую станцию, универсальный компьютер, персональный компьютер, мобильное устройство и т.д. Секвенатор нуклеиновых кислот может быть настроен на анализ (например, распознавание) фрагмента нуклеиновой кислоты (например, одного фрагмента, фрагментов-партнеров, фрагмента со спаренными концами и т.д.) с использованием всего доступного разнообразия способов, платформ или технологий для получения информации последовательности нуклеиновой кислоты, в частности, способов, описанных в настоящей заявке с использованием композиций, предложенных в настоящей заявке. Согласно некоторым вариантам реализации изобретения, секвенатор нуклеиновых кислот сообщается с накопителем данных образца последовательности прямо через кабель данных (например, последовательный кабель, прямое кабельное сообщение и т.д.) или шинное соединение или, альтернативно, через сетевое соединение (например, интернет, LAN, WAN, VPN и т.д.). Согласно некоторым вариантам реализации изобретения, сетевое соединение может представлять собой «жесткое» механическое соединение. Например, секвенатор нуклеиновых кислот может быть коммуникативно связан (через Category 5 (САТ5), оптоволокно или эквивалентную кабельную сеть) с данными сервера, то есть коммуникативно связан (через САТ5, оптоволокно или эквивалентную кабельную сеть) через интернет и накопителем данных образца последовательности. Согласно некоторым вариантам реализации изобретения, сетевое соединение представляет собой беспроводное сетевое соединение (например, Wi-Fi, WLAN и т.д.), например, использующее 802,11 a/b/g/n или эквивалентный формат передачи данных. На практике, используемое сетевое соединение зависит от конкретных требований системы. Согласно некоторым вариантам реализации изобретения, накопитель данных образца последовательности представляет собой интегрированный компонент секвенатора нуклеиновых кислот.
Согласно некоторым вариантам реализации изобретения, накопитель данных образца последовательности представляет собой любое устройство хранения базы данных, систему или обеспечение (например, раздел накопителя данных и т.д.) настроенное на организацию и хранение данных прочтения последовательности нуклеиновой кислоты, генерированных секвенатором нуклеиновых кислот таким образом, что указанные данные можно искать и извлекать вручную (например, с помощью администратора базы данных или оператора-клиента) или автоматически с помощью компьютерной программы, приложения или программного сценария. Согласно некоторым вариантам реализации изобретения, накопитель референсных данных может представлять собой любое устройство, накопительную систему или обеспечение базы данных (например, раздел накопителя данных и т.д.) настроенный на организацию и хранение референсных последовательностей (например, целого генома или части генома, целого экзома или части экзома, SNP, гена и т.д.) таким образом, что данные можно искать и извлекать вручную (например, с помощью администратора или оператора-клиента базы данных) или автоматически с помощью компьютерной программы, приложения и/или программного сценария. Согласно некоторым вариантам реализации изобретения, данные секвенирования прочтения образца нуклеиновой кислоты можно хранить на накопителе данных образца последовательности и/или накопителе референсных данных в виде файлов различных типов/форматов разных данных, включая, но не ограничиваясь указанными: *.txt, *.fasta, *.csfasta, *seq.txt, *qseq.txt, *.fastq, *.sff, *prb.txt, *.sms, *srs и/или *.qv.
Согласно некоторым вариантам реализации изобретения, накопитель данных образца последовательности и накопитель референсных данных представляют собой независимые автономные устройства/системы или реализуются на разных устройствах. Согласно некоторым вариантам реализации изобретения, накопитель данных образца последовательности и накопитель референсных данных реализуются на одном и том же устройстве/системе. Согласно некоторым вариантам реализации изобретения, накопитель данных образца последовательности и/или накопитель референсных данных может реализоваться на аналитическом вычислительном устройстве/сервере/узле. Аналитическое вычислительное устройство/сервер/узел может сообщаться с накопителем данных образца последовательности и накопителем референсных данных прямо через кабель данных (например, последовательный кабель, прямое кабельное соединение и т.д.) или шинное соединение или, альтернативно, через сетевое соединение (например, Интернет, LAN, WAN, VPN и т.д.). Согласно некоторым вариантам реализации изобретения, аналитическое вычислительное устройство/сервер/узел может содержать механизм референсного картирования, модуль de novo картирования и/или механизм третичного анализа. Согласно некоторым вариантам реализации изобретения, механизм референсного картирования может быть настроен на получение прочтений образца последовательности нуклеиновой кислоты из накопителя данных образца и картировать их к одной или более референсным последовательностям, полученным из накопителя референсных данных для сборки прочтений в последовательность, которая подобна, но не обязательно идентична референсной последовательности, с использованием всего разнообразия техник и способов референсного картирования/выравнивания. Заново собранную последовательность затем можно дополнительно анализировать с помощью одного или более необязательных механизмов третичного анализа для идентификации различий в генетической структуре (генотипе), экспрессии гена или эпигенетическом статусе индивида, которые могут приводить к большим различиям в физических характеристиках (фенотипе). Например, согласно некоторым вариантам реализации изобретения, механизм третичного анализа может быть настроен на идентификацию различных геномных вариантов (в собранной последовательности) из-за мутаций, рекомбинации/кроссинговера или дрейфа генов. Примеры типов геномных вариантов включают, но не ограничиваются указанными: однонуклеотидные полиморфизмы (SNP), вариации количества копий (CNV), инсерции/делеции (инсерционно-делеционные мутации), инверсии и т.д. Необязательно модуль de novo картирования может быть настроен на сборку прочтений образца последовательности нуклеиновой кислоты из накопителя данных образца в новые и ранее не известные последовательности. Необходимо понимать, однако, что различные механизмы и модули, размещенные на аналитическом вычислительном устройстве/сервере/узле, могут быть объединены или сжаты в один механизм или модуль в зависимости от требований конкретного приложения или архитектуры системы. Более того, согласно некоторым вариантам реализации изобретения, аналитическое вычислительное устройство/сервер/узел может содержать дополнительные механизмы или модули, в соответствии с требованиями конкретных приложений или архитектуры системы.
Согласно некоторым вариантам реализации изобретения, механизмы картирования и/или третичного анализа настраивают на обработку прочтений нуклеиновой кислоты и/или референсной последовательности в цветовом пространстве. Согласно некоторым вариантам реализации изобретения, механизмы картирования и/или третичного анализа настраивают на обработку прочтений нуклеиновой кислоты и/или референсной последовательности в базисном пространстве. Необходимо понимать, однако, что механизмы картирования и/или третичного анализа, описанные в настоящей заявке, могут обеспечивать обработку или анализ последовательности нуклеиновой кислоты, приведенной в виде любой схемы или в любом формате, при условии что указанная схема или формат может передавать идентичность оснований и положений последовательности нуклеиновой кислоты.
Согласно некоторым вариантам реализации изобретения, данные прочтения последовательности образца нуклеиновой кислоты и референсной последовательности могут доставляется в аналитическое вычислительное устройство/сервер/узел в виде файлов входных данных различных типов/форматов, включая, но не ограничиваясь указанными: *.txt, *.fasta, *.csfasta, *seq.txt, *qseq.txt, *.fastq, *.sff, *prb.txt, *.sms, *srs и/или *.qv.
Более того, клиентский терминал может представлять собой «тонкое» или «толстое» клиентское вычислительное устройство. Согласно некоторым вариантам реализации изобретения, клиентский терминал может содержать интернет-браузер, который может использоваться для контроля работы механизма референсного картирования, модуля de novo картирования и/или механизма третичного анализа. То есть клиентский терминал может иметь доступ к механизму референсного картирования, модулю de novo картирования и/или механизму третичного анализа с использованием браузера для контроля их функции. Например, клиентский терминал можно использовать для конфигурации рабочих параметров (например, лимит ошибок, порогового значения балльной оценки качества и т.д.) различных механизмов, в зависимости от требований конкретного приложения. Подобным образом, клиентский терминал также может воспроизводить результаты анализа, полученные с помощью механизма референсного картирования, модуля de novo картирования и/или механизма третичного анализа.
Технологии согласно настоящему изобретению также включают любой способ, способный получать, обрабатывать и передавать информацию лабораториям, проводящим анализы, провайдерам информации, медицинскому персоналу и субъектам, и от них.
9. Способы применения
Технология не ограничивается конкретными способами применения, а применяется в широком диапазоне исследовательских (фундаментальных и прикладных), клинических, медицинских и других биологических, биохимических и молекулярно-биологических применений. Некоторые типичные способы применения технологии включают область генетики, геномики и/или генотипирования, например, растений, животных и других организмов, например, для идентификации гаплотипов, фазирования и/или сцепления мутаций и/или аллелей. Конкретные и неограничивающие иллюстративные примеры в области медицины для человека включают исследование на муковисцидоз и синдром ломкой Х-хромосомы.
Кроме того, технология применяется в области инфекционных заболеваний, например, для идентификации инфекционных агентов, таких как вирусы, бактерии, грибы и т.д., и для определения типов, семейств, видов и/или квазивидов вирусов и для идентификации гаплотипов, фазирования и/или сцепления мутаций и/или аллелей. Конкретный и неограничивающий иллюстративный пример в области инфекционных заболеваний представляет собой характеристику генетических элементов вируса иммунодефицита человека (ВИЧ) и идентификацию гаплотипов, фазирования и/или сцепления мутаций и/или аллелей. Другие конкретные и неограничивающие иллюстративные примеры в области инфекционных заболеваний включают характеристику детерминант устойчивости к антибиотикам; отслеживание инфекционных организмов для эпидемиологии; мониторинг возникновения и эволюции механизмов устойчивости; идентификацию видов, подтипов, цепей, экстрахромосомных элементов, типов и т.д., связанных с вирулентностью, мониторинг прогресса лечения и т.д.
Согласно некоторым вариантам реализации изобретения, технология применяется в трансплантационной медицине, например, для типирования главного комплекса гистосовместимости (МНС), типирования человеческого лейкоцитарного антигена (HLA) и для идентификации гаплотипов, фазирования и/или сцепления мутаций и/или аллелей, связанный с трансплантационной медициной (например, для идентификации совместимых доноров для конкретного хозяина, нуждающегося в трансплантации, для предсказания риска отторжения, для мониторинга отторжения, для хранения трансплантационного материала, для создания баз данных медицинской информатики и т.д.).
Согласно некоторым вариантам реализации изобретения, технология применяется в онкологии и областях, связанных с онкологией. Конкретные и неограничивающие иллюстративные примеры в области онкологии представляют собой идентификацию генетических и/или геномных аббераций, связанных с раком, предрасположенностью к раку и/или лечением рака. Например, согласно некоторым вариантам реализации изобретения, технология применяется в выявлении наличия хромосомной транслокации, связанной с раком; и согласно некоторым вариантам реализации изобретения, технология применяется в идентификации новых партнеров слияния генов для получения диагностических тестов для выявления рака. Согласно некоторым вариантам реализации изобретения, технология применяется в скрининге раковых заболеваний, диагностике раковых заболеваний, прогнозе ракового заболевания, измерении минимального остаточного заболевания и выборе и/или мониторинге курса лечения рака.
Согласно некоторым вариантам реализации изобретения, технология применяется для характеризации нуклеотидных последовательностей. Например, согласно некоторым вариантам реализации изобретения, технология применяется для выявлении инсерций и/или делеций («инсерционно-делеционных мутаций») в нуклеотидной последовательности (например, геноме, гене и т.д.). Предполагается, что технология, описанная в настоящей заявке, обеспечивает улучшенное выявление инсерционно-делеционных мутаций по сравнению со стандартными технологиями. Кроме того, технология применяется для выявления коротких тандемных повторов (STR), инверсий, больших инсерций и для секвенирования повторяющихся (например, высоко повторяющихся) областей нуклеотидной последовательности (например, генома).
Несмотря на то что изобретение, описанное в настоящей заявке, относится к конкретным показанным вариантам реализации, необходимо понимать, что указанные варианты реализации приведены в качестве примера и не ограничивают настоящее изобретение.
Примеры
Пример 1 - сравнение с системой Illumina MiSeq
В процессе разработки технологии, предложенной в настоящей заявке, проводили расчеты для сравнения производительности технологии, предложенной в настоящей заявке (Таблицы 1 и 2, «библиотека SOD») и стандартной технологии, предложенной компанией Illumina на платформе MiSeq (Таблицы 1 и 2, «библиотека ампликонов Illumina»). Собирали данные для двух различающихся сценариев, например, количество образцов на рабочий цикл, критерии для измерения производительности и т.д. (см. Таблицы 1 и 2).
Как показано в Таблицах 1 и 2, технология, описанная в настоящей заявке, обеспечивает снижение инструментального времени выполнения, имеет более высокую производительность и обеспечивает получение более высокого процента прочтений со значением балльной оценки качества более чем Q30 по сравнению с созданием библиотеки NGS с использованием технологии Illumina.
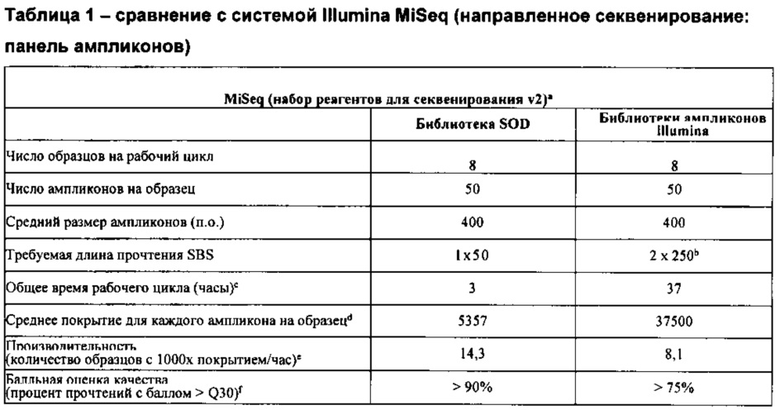
a) Набор реагентов MiSeq v2: двустороннее сканирование, 12-15 миллионов отфильтрованных кластеров
b) Для покрытия целого ампликона размером 400 п.о. использовали стратегию прочтений со спаренными концами размером 2×250 п.о., где указанные прочтения перекрываются на ~100 п.о.
c) Реальное время, затраченное только на секвенирование (не включает время образования кластеров)
d) Для расчета покрытия для библиотеки SOD: [(Общее # прочтений)/((размер вставки - длина прочтения SOD) × (# образцов за рабочий цикл × # ампликонов на образец))] × длина прочтения SOD: например, [(15×106)/((400-50)×(8×50))]×50
e) Для расчета производительности: [(среднее покрытие)/1000]/(общее время рабочий цикла)
f) На основе инструкции для секвенирования с помощью системы MiSeq, предложенной компаниейу Illumina, например, в их доступных в интернете материалах.

a) Набор реагентов MiSeq v2: двустороннее сканирование, 15 миллионов отфильтрованных кластеров
b) Для покрытия целого ампликона размером 200 или 400 п.о. использовали стратегию прочтений со спаренными концами размером 2×150 или 2×250 п.о. (соответственно), где прочтения перекрываются на ~100 п.о.
c) Реальное время, затраченное только на секвенирование (не включает время образования кластеров)
d) Для расчета покрытия для библиотеки SOD: [(Общее # прочтений)/((размер вставки - длина прочтения SOD) × (# образцов на рабочий цикл × # ампликонов на образец))] × длина прочтения SOD: например, [(15×106)/((400-50)×(8×50))]×50
e) Для расчета производительности: [(среднее покрытие)/2000]/(общее время рабочий цикла)
f) На основе инструкции для секвенирования с помощью MiSeq, предложенной компанией Illumina, например, в их материалах, доступных в интернете.
Пример 2 - сравнение с системой PGM, Ion Torrent (направленное секвенирование: панель ампликонов)
В процессе разработки технологии, предложенной в настоящей заявке, проводили расчеты для сравнения производительности технологии, предложенной в настоящей заявке (Таблицы 3 и 4, «библиотека SOD»), и стандартной технологии, предложенной компанией Ion Torrent на платформе PGM (Таблицы 3 и 4, «библиотека ампликонов Ion»). Собирали данные для двух отличающихся сценариев, например, количество образцов на рабочий цикл, критерий для измерения производительности и т.д. (см. Таблицы 3 и 4).
Как показано в Таблицах 3 и 4, технология, описанная в настоящей заявке, обеспечивает снижение инструментального времени выполнения и приводит к получению более высокого процентного содержания прочтений с балльной оценкой качества более чем Q20 по сравнению с созданием библиотеки NGS с использованием технологии Ion Torrent.
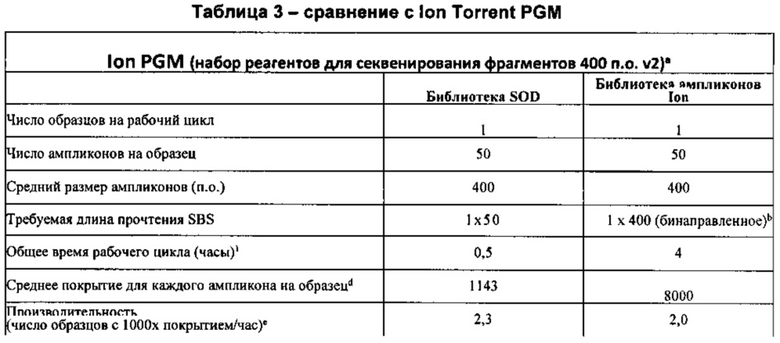

a) Набор реагентов для секвенирования 400 п.о. PGM v2
b) Для покрытия целого ампликона размером 400 п.о. осуществляли бинаправленное секвенирование 1×400 п.о.
c) Реальное время, затраченное только на секвенирование (не включает OneTouch2 и время других предшествующих секвенированию процессов)
d) Для расчета покрытия для библиотеки SOD: [(0,4×106)/((400-50)×(8×50))]×50
e) Для расчета производительности: [(среднее покрытие)/1000]/(общее время рабочего цикла)
f) Спроектировано на основе: Loman N. et al. (2012) "Performance comparison of benchtop high-throughput sequencing platforms" Nature Biotechnology; vol. 30-5
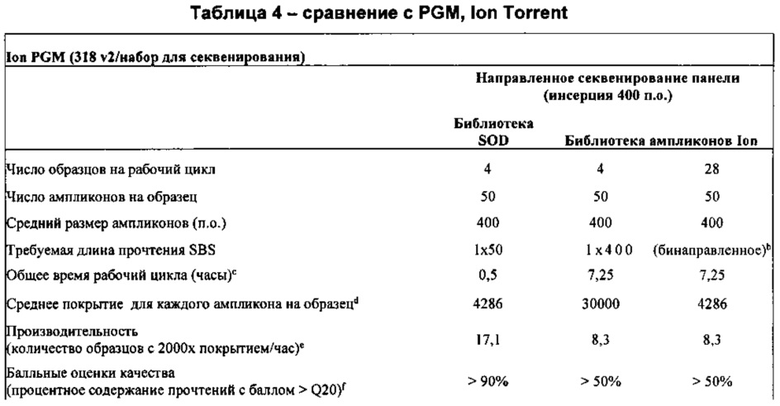
a) Чип Ion PGM 318/v2: лунки с нагрузкой ~ 6 миллионов, приводящие к получению отфильтрованных прочтений
b) Для покрытия целого ампликона размером 200 п.о. или 400 п.о. осуществляют стратегию прочтений размером 200 п.о. (бинаправленное секвенирование) или 400 п.о. (бинаправленное секвенирование)
c) Реальное время, затраченное только на секвенирование (не включает эПЦР/обогащение)
d) Для расчета покрытия для библиотеки SOD: [(#общих прочтений)/((размер вставки - длина прочтения SOD) × (# образцов × # ампликонов))] × длина прочтения SOD, например, [(15×106)/((400-50)×(8×50))]×50
e) Для расчета производительности: [(среднее покрытие)/2000]/(общее время рабочего цикла)
f) На основе инструкции для секвенирвоания Ion Torrent, доступной в интернет-материалах Ion Torrent
Пример 3 - Сравнение технологий для длинных прочтений
В Таблицах 5 и 6 приведено сравнение производительности технологий, предложенных в настоящей заявке, со стандартными технологиями секвенирования длинных ампликонов, состоящих приблизительно из 1000 п.о. (Таблица 5) и 2000 п.о. (Таблица 6). Время выполнения не повышается с увеличением размера ампликона при технологии согласно настоящему изобретению, так как размер прочтения составляет ~30-50 оснований, независимо от размера нуклеиновой кислоты-мишени, подлежащей секвенированию. Согласно некоторым вариантам реализации изобретения, последовательность из 2000 п.о. получают с помощью технологии, предложенной в настоящей заявке, за время, порядок величины которого меньше по сравнению со стандартной технологией (см., например, Таблицу 6). Согласно некоторым вариантам реализации изобретения, технология, предложенная в настоящей заявке, обеспечивает более длинное прочтение последовательности при таком же времени рабочего цикла, как при стандартной технологии.
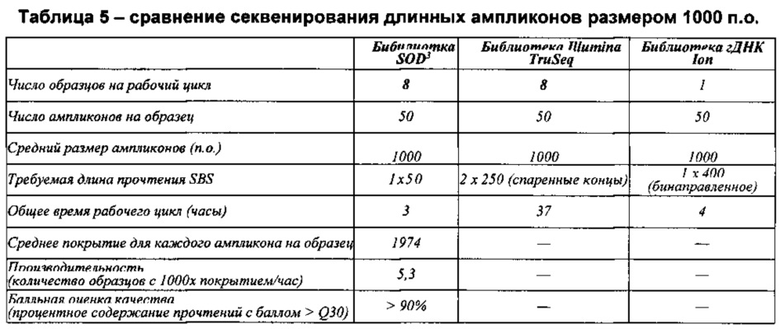
а) Рабочий цикл библиотеки SOD на приборе MiSeq с набором реагента секвенирования v2
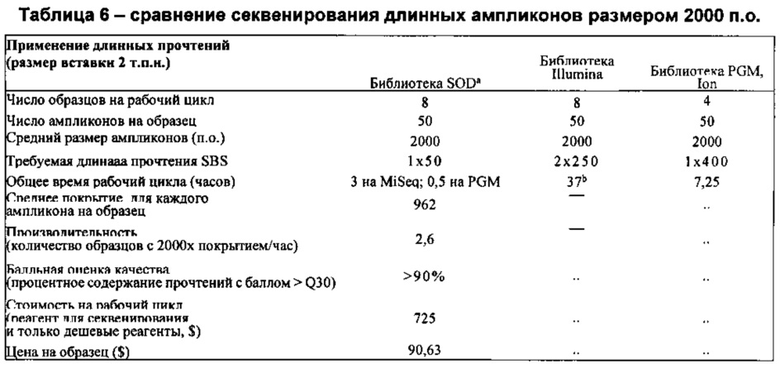
a) время получения библиотеки SOD для более длинной вставки больше согласно некоторым вариантам реализации изобретения (например, от ~6,5 часов до ~8,5 часов)
b) технология Illumina «Moleculo»
Пример 4 - Концептуальная проверка данных, полученных с использованием модельной библиотеки
В процессе разработки вариантов реализации технологии, предложенных в настоящей заявке, собирали данные для проверки технологии с использованием модельной библиотеки. Как показано на Фигуре 4, консенсусная последовательность размером ~127 п.о. сконструирована из коллекции прочтений размером ~35 п.о., полученных в соответствии с предложенными вариантами реализации технологии. Рассчитанное время рабочего цикла секвенирования на приборе для секвенирования ДНК Illumina MiSeq для получения последовательности размером ~127 п.о. с использованием библиотеки, полученной с помощью технологии, предложенной в настоящей заявке, составляло приблизительно 2,5 часа. При использовании стандартной технологии получения библиотеки время рабочего цикла, составляющее ~13 часов, приводило к получению такого же прочтения последовательности размером ~127 п.о.
Пример 5 - создание «лестницы» с использованием терминации с помощью 3'-О-пропаргил dNTP
В процессе разработки вариантов реализации технологии, представленной в настоящей заявке, проводили эксперименты для оценки создания терминированных фрагментов нуклеиновой кислоты в реакции, содержащий смесь 3'-О-пропаргил-dNTP и природные (стандартные) dNTP. В частности, проводили эксперименты для оценки создания фрагментов, терминированных по каждому положению в области-мишени путем включения терминирующих цепь 3'-О-пропаргил-dNTP с помощью ДНК-полимеразы во время синтеза. Анализы полимеразного удлинения проводили с использованием матрицы нуклеиновой кислоты, содержащей последовательность из человеческого гена KRAS (например, экзона 2 гена KRAS и фланкирующих интронных последовательностей) и комплементарный праймер:
Матрица экзона 2 гена KRAS (SEQ ID NO: 1)

R_ke2_trP1_T_bio (SEQ ID NO: 2)
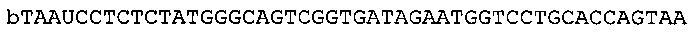
В последовательности праймера R_ke2_trP1_T_bio (SEQ ID NO: 2) «b» показывает биотиновую модификацию, и «U» показывает дезоксиуридиновую модификацию. Включение праймеров в продукты удлинения приводит к получению продуктов удлинения, содержащих урацил. Урацил применим, например, для расщепления продукта (например, с использованием реагентов расщепления урацила) при некоторых молекулярно-биологических манипуляциях (например, отщеплении продукта от твердой подложки).
Эксперименты проводили с использованием смеси природных dNTP и всех четырех из 3'-О-пропаргил-dNTP в одной реакции. Получение реакционной смеси фрагментов ДНК включало 20 мМ Tris-HCl, 10 мМ (NH4)SO4, 10 мМ KCl, 2 мМ MnCl2, 0,1% Triton Х-100, 1000 пкмоль dATP, 1000 пкмоль dCTP, 1000 пкмоль dGTP, 1000 пкмоль dTTP, 100 пкмоль 3'-О-пропаргил-dATP, 100 пкмоль 3'-О-пропаргил-dCTP, 100 пкмоль 3'-О-пропаргил-dGTP, 100 пкмоль 3'-О-пропаргил-dTTP, 6,25 пкмоль праймера R_ke2_trP1_T_bio (SEQ ID NO=2) и 2 единицы ДНК-полимеразы THERMINATOR II (New England BioLabs) в реакционном объеме 25 мкл. 0,5 пкмоль очищенного ампликона, соответствующего области в экзоне 2 гена KRAS (SEQ ID NO=1) использовали в качестве матрицы. Термоциклирование реакции полимеразного удлинения проводили путем нагрева до 95°С в течение 2 минут с последующими 45 циклами при 95°С в течение 15 секунд, при 55°С в течение 25 секунд и при 65°С в течение 35 секунд.
После реакции полимеразного удлинения 1 мкл реакционной смеси использовали непосредственно для анализа размера ДНК-фрагмента с использованием гель-электрофореза (Agilent 2100 Bioanalyzer и высокочувствительный чип для анализа ДНК). Анализ размера фрагментов продуктов реакции показал, что реакция создания фрагментов приводила к успешному получению «лестницы» фрагментов нуклеиновой кислоты, имеющих ожидаемые размеры.
Пример 6 - синтез 5'-азидометил-модифицированного олигонуклеотида
В процессе разработки вариантов реализации технологии, предложенной в настоящей заявке олигонуклеотид, содержащий 5'-азидометил-модификацию, синтезировали и охарактеризовали. Синтез модифицированного олигонуклеотида осуществляли с использованием фосфорамидитной химической синтетической реакции. В последнем этапе синтеза использовали фосфорамидитную химическую синтетическую реакцию для включения 5'-йод-dT фосфорамидита в концевое 5' положение. Олигонуклеотид, присоединенный к твердой подложке в реакционной колонке, затем обрабатывали, как следует далее.
Сначала азид натрия (30 мг) ресуспендировали в безводном ДМФА (1 мл), нагревали в течение 3 часов при 55°С и охлаждали до комнатной температуры. Супернатант отбирали с помощью шприца на 1 мл и пропускали туда и обратно через реакционную колонку, содержащую 5'-йод-модифицированный олигонуклеотид, и инкубировали в течение ночи при температуре окружающей среды (при комнатной температуре). После инкубации колонку промывали безводным ДМФА, ацетонитрилом и затем сушили с помощью газообразного аргона. Полученный 5'-азидометил-модифицированный олигонуклеотид отщепляли от твердой подложки и защитную группу удаляли путем нагревания в водном аммиаке в течение 5 часов при 55°С. Конечный продукт представлял собой олигонуклеотид, имеющий последовательность, показанную ниже:
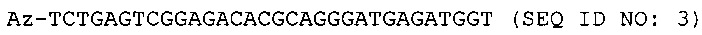
«Az» указывает на азидометильную модификацию на 5' конце (например, 5'-азидо-метил-модификацию), например, для получения олигонуклеотида, содержащего структуру, соответствующую следующей структуре:
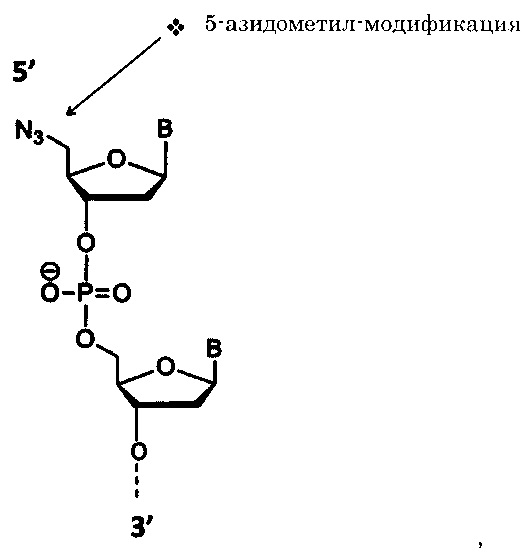
где В представляет собой основание нуклеотида (например, аденин, гуанин, тимин, цитозин или природное или синтетические нуклеиновое основание, например, модифицированный пурин, такой как гипоксантин, ксантин, 7-метилгуанин; модифицированный пиримидин, такой как 5,6-дигидроурацил, 5-метилцитозин, 5-гидроксиметилцитозин и т.д.).
Пример 7 - конъюгирование 5-азидо-метил-модифицированного олигонуклеотида и 3'-О-пропаргил-модифицированных фрагментов нуклеиновой кислоты
В процессе разработки вариантов реализации технологии, предложенной в настоящей заявке, проводили эксперименты для анализа конъюгирования 5'-азидо-метил-модифицированного олигонуклеотида (например, см. Пример 6) с 3'-О-пропаргил-модифицированными фрагментами нуклеиновой кислоты (например, см. Пример 5) с помощью реакции «клик-химии». В частности, проводили эксперименты, в которых 5'-азидо-метил-модифицированный олигонуклеотид подвергали химическому конъюгированию с 3'-О-пропаргил модифицированными фрагментами ДНК с использованием химической реакции 1,3-биполярного азид алкинового циклоприсоединения, катализируемого медью (I) (реакции «клик-химии»).
Реакцию «клик-химии» осуществляли с использованием коммерчески доступных реагентов (Baseclick GmbH, набор Олиго-Click-M Reload) в соответствии с инструкциями производителя. Вкратце, приблизительно 0,1 пкмоль 3'-О-пропаргил-модифицированных фрагментов ДНК, содержащих 5'-биотиновую модификацию, подвергали реагированию приблизительно с 500 пкмоль 5'-азидо-метил-модифицированного олигонуклеотида с использованием реагента реакции «клик-химии» в общем объеме, составляющем 10 мкл. Реакционную смесь инкубировали при 45°С в течение 30 минут.После инкубирования супернатант переносили в новую микроцентрифужную пробирку и добавляли коммерческий связывающий и промывочный буфер в объеме 40 мкл (например, 1 M NaCl, 10 мМ Tris-HCl, 1 мМ EDTA, рН 7,5). Конъюгированный продукт реакции выделяли из избыточного количества 5'-азидо-метил-модифицированного олигонуклеотида путем инкубации смеси для реакции «клик-химии» с покрытыми стрептавидином магнитными гранулами (Dynabeads, MyOne Streptavidin C1, Life Technologies) при температуре окружающей среды (комнатной температуре) в течение 15 минут. Гранулы отделяли от супернатанта с помощью магнита и супернатант удаляли. Затем гранулы промывали два раза с использованием связывающего и промывочного буфера и ресуспендировали в 25 мкл буфера ТЕ (10 мМ Tris-HCl, 0,1 мМ EDTA, рН приблизительно 8).
Продукт отщепляли от твердого носителя (гранулы) путем расщепления урацила (с помощью урацилгликозилазы и эндонуклеазы VIH, Enzymatics). В частности, реагенты расщепления урацила использовали для расщепления продуктов реакции по сайту дезоксиуридиновой модификации, расположенной около 5'-конца конъюгированного продукта (см. SEQ ID NO: 2-5). В конечном итоге супернатант, содержащий конъюгированный продукт, очищали с использованием системы Ampure ХР (Beckman Coulter) в соответствии с протоколом производителя и элюировали в 20 мкл буфера ТЕ.
Пример 8 - амплификация конъюгированного продукта
В процессе разработки вариантов реализации технологии, описанной в настоящей заявке, проводили эксперименты для характеризации химического конъюгирования 5'-азидометил-модифицированного олигонуклеотида с 3'-О-пропаргил-модифицированными фрагментами нуклеиновой кислоты и для оценки триазольной связи, имитирующей природную фосфодиэфирную связь в скелете нуклеиновой кислоты. Для исследования способности полимеразы распознавать конъюгированный продукт как матрицу и вращать триазольную связь во время синтеза были сконструированы праймеры для ПЦР с получением ампликонов, которые соединяют триазольные связи продуктов конъюгирования:
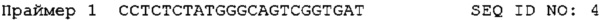
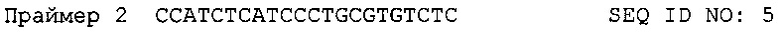
Коммерчески доступную готовую смесь для ПЦР (KAPA 2G HS, KAPA Biosystems) использовали для получения 25 мкл реакционной смеси, содержащей помимо компонентов, представленных в готовой смеси (например, буфера, полимеразы, dNTP), 0,25 мкМ праймера 1 (SEQ ID NO: 4), 0,25 мкМ праймера 2 (SEQ ID NO: 5) и 2 мкл очищенного конъюгированного продукта (см. Пример 7) в качестве матрицы для амплификации. Термоциклирование реакционной смеси проводили путем инкубации образца при 95°С в течение 5 минут с последующими 30 циклами при 98°С в течение 20 секунд, при 60°С в течение 30 секунд и при 72°С в течение 20 секунд. Продукты амплификации анализировали с помощью электрофореза в геле (например, с использованием системы Agilent Bioanalyzer 2100 и высокочувствительного ДНК-чипа) для определения распределения продуктов реакции по размеру.
Анализ продуктов амплификации показал, что реакция амплификации приводила к успешному получению ампликонов с использованием конъюгированных продуктов реакции «клик-химии» (см. Пример 7) в качестве матриц для амплификации. В частности, анализ продуктов амплификации показал, что полимераза функционировала на протяжении матрицы через триазольную связь с получением ампликонов на основе указанной матрицы. Кроме того, амплификация приводила к получению гетерогенной популяции ампликонов, имеющих диапазон размеров, соответствующих ожидаемым размерам, полученным в результате амплификации терминированных специфично в отношении оснований фрагментов ДНК путем включения 3'-О-пропаргил-dNTP. Анализ фрагмента также показал подходящее увеличение размера фрагмента, соответствующее тридцать одному (31) дополнительному основанию из конъюгированного 5'-азидометил-модифицированного олигонуклеотида.
Пример 9 - лигирование адаптеров NGS с продуктами «лестницы» фрагментов
В процессе разработки вариантов реализации технологии, предложенной в настоящей заявке проводили эксперименты для секвенирования «лестницы» фрагментов, полученных согласно технологии, предложенной в настоящей заявке (см. Фигура 5). В качестве исходного этапа секвенирования эксперименты проводили для получения библиотеки секвенирования с использованием продуктов «лестницы» ДНК, полученных в Примере 8, в качестве входных данных и коммерческого набора для получения образца. Библиотеки секвенирования получали с использованием набора для получения образцов ДНК TRUSEQ NANO (Illumina, Inc.) в соответствии с протоколом производителя со следующей модификацией. После этапа лигирования адаптера проводили два цикла (вместо одного цикла) очистки на основе гранул с использованием отношения 1:1 (по объему) образца к смеси гранул. 8 циклов амплификации проводили с использованием предложенных Illumina праймеров для ПЦР для обогащения лигированных с адаптером продуктов в соответствии с протоколом производителя. Конечную библиотеку секвенирования анализировали с помощью электрофореза в геле (Agilent 2100 Bioanalyzer и высокочувствительного чипа для анализа ДНК). Анализ размеров фрагментов подтвердил успешное получение библиотеки NGS (например, для секвенирования с помощью Illumina) с использованием продуктов «лестницы» фрагментов Примера 8. Данные указывали на то, что в библиотеке NGS наблюдается подходящее увеличение размера фрагментов, соответствующее добавлению адаптеров размером 126 п.о. фирмы Illumina и, таким образом, и что адаптеры были советующим образом лигированы с фрагментами «лестницы». На Фигуре 5 показана схему фрагментов библиотеки для секвенирования. В частности, фрагменты включают адаптер Issumina на обоих концах, одну или более универсальных последовательностей и последовательность-мишень.
Пример 10 - секвенирование
В процессе разработки вариантов реализации технологии, предложенной в настоящей заявке, проводили эксперименты для секвенирования лигированной с адаптером библиотеки NGS, например, библиотеки для секвенирования, полученной, как описано в Примере 9. Библиотеку, полученную в соответствии с Примером 9, успешно секвенировали с использованием секвенатора MiSeq фирмы Illumina с использованием набора для секвенирования последовательностей размером 2×75 п.о. путем синтеза. Праймеры для секвенирования, комплементарные последовательностям адаптеров, предложены в наборе. После секвенирования более чем 89% прочтений имели балльную оценку качества последовательности Q30 или лучше.
Данные, полученные в результате экспериментов, показали, что популяция фрагментов обеспечивает точное выравнивание коротких прочтений секвенирования (30-50 п.о.), полученных с помощью технологии. В частности, перекрывающиеся фрагменты нуклеиновой кислоты обеспечивали прочтения, которые успешно выравнивались и собирались, несмотря на их малый размер.
Данные последовательности извлекали из выходных данных секвенатора с использованием индивидуального производственного процесса обработки данных, который обеспечивает конкретный дизайн фрагмента «лестницы» фрагментов, созданной согласно технологии. Например, индивидуальное программное обеспечение обеспечивало идентификацию и обработку прочтений для использования частей размером 40 п.о. прочтений последовательности 2×75 п.о. для последующего выравнивания последовательностей. Конкретные компоненты индивидуального программного обеспечения обеспечивают соединение прочтений (например, файлов FASTQ прочтения 1 и прочтения 2), полученных из секвенатора NGS; идентификацию последовательности, происходящей из последовательности-мишени, универсальной последовательности и адаптеров (например, идентификацию последовательности, происходящей из 5'-азидометил-олигонуклеотида); установку границы извлечения последовательности с помощью распознавания структур; выделение последовательности-мишень из прочтений последовательности, полученных из секвенатора NGS, и выравнивание последовательностей (см. Фигуру 5).
Пример 11 - выравнивание последовательности
В процессе разработки вариантов реализации технологии, предложенной в настоящей заявке, проводили эксперименты для выравнивания данных последовательности, полученных из библиотеки NGS, как описано в настоящей заявке, для получения консенсусной последовательности в результате выравнивания и выравнивания консенсусной последовательности по отношению к референсной последовательности. В частности, прочтения последовательности, содержащие 40 п.о., которые были выделены из выходных данных секвенирования с помощью MiSeq, выравнивали по отношению к референсной последовательности (например, последовательности, содержащей 177 п.о., содержащей экзон 2 человеческого гена KRAS, частично фланкирующей интронные последовательности).
Выравнивание прочтений секвенирования, содержащих 40 п.о., осуществляли с использованием системы CLC, Genomics Workbench v7 со строгими «штрафами» за ошибки и инсерционно-делеционные мутации; требования относительно совпадения длины и подобия подходящим образом устанавливались согласно сопутствующей инструкции для прочтений, содержащих 40 п.о. Результаты выравнивания (Фигура 6А) показали, что прочтения последовательности, содержащие 40 п.о., обеспечивали полное покрытие целой референсной последовательности (177 т.п.н.). Кроме того, график глубины покрытия в зависимости от положения последовательности отражал ожидаемый «трапециевидный» профиль покрытия, который был установлен в ходе теоретического моделирования выравнивания (Фигура 6В).
Описанные результаты указывают на то, что относительно короткий рабочий цикл секвенатора (например, MiSeq с 30-50 циклами секвенирования путем синтеза) приводит к получению полной последовательности-мишени высокого качества. Кроме того, можно максимально увеличить длину последовательности высокого качества с помощью корректировки существующих способов, например, путем создания праймеров, связывающихся непосредственно рядом с сайтом-мишенью. Кроме того, длину последовательности высокого качества также можно максимально увеличить при создании подходящей «лестницы» фрагментов, покрывающих всю длину полноразмерной мишени (например, путем регулирования отношения 3'-О-пропаргил-dNTP к dNTP; см. Пример 12). В указанном примере 40 циклов секвенирования (для получения 40 оснований последовательности) на MiSeq занимали приблизительно 2,5 часов. Важно, что технология обеспечивает преимущество по сравнению с существующими технологиями, которое заключается в том, что время работы секвенатора не изменяется в зависимости от размера мишени.
Пример 12 - секвенирование и анализ библиотек NGS
В процессе разработки вариантов реализации технологии, предложенной в настоящей заявке, проводили эксперименты для контроля распределения по размеру терминированных фрагментов нуклеиновой кислоты, полученных в результате реакции, содержащей смесь 3'-О-пропаргил-dNTP и природных (стандартных) dNTP, путем регулирования отношения 3'-О-пропаргил-dNTP к природным (стандартных) dNTP. Предполагалось, что молярное отношение 3'-О-пропаргил-dNTP к природным dNTP влияет на распределение фрагментов по размеру в связи с конкуренцией между 3'-О-пропаргил-dNTP (который терминирует удлинение) и природными dNTP (которые удлиняют полимеразные продукты) за включение в синтезируемую нуклеиновую кислоту с помощью полимеразы.
Соответственно, проводили эксперименты, в которых продукты реакций создания «лестницы» фрагментов оценивали при различных молярных отношениях 3'-О-пропаргил-dNTP к природным dNTP. Реакции создания «лестницы» фрагментов проводили с использованием молярных отношений 2:1, 10:1 и 100:1 природных dNTP к 3'-О-пропаргил-dNTP. Реакционные смеси для создания фрагментов, используемых в указанных экспериментах, включали 20 мМ Tris-HCl, 10 мМ (NH4)SO4, 10 мМ KCl, 2 мМ MnCl2, 0,1% Triton Х-100, 1000 пкмоль dATP, 1000 пкмоль dCTP, 1000 пкмоль dGTP, 1000 пкмоль dTTP, 6,25 пкмоль праймера, 2 единицы ДНК-полимеразы Therminator II (New England BioLabs) и 0,5 пкмоль очищенного ампликона, соответсвующего области в экзоне 2 гена KRAS (SEQ ID NO: l), в качестве матрицы в конечном реакционном объеме 25 мкл.
Кроме того, реакции, анализирующие отношение природных dNTP к 3'-О-пропаргил-dNTP 2:1, включали 500 пкмоль 3'-О-пропаргил-dATP, 500 пкмоль 3'-O-пропаргил-dCTP, 500 пкмоль 3'-О-пропаргил-dGTP и 500 пкмоль 3'-О-пропаргил-dTTP. Реакции, анализирующие отношение природных dNTP к 3'-О-пропаргил-dNTP 10:1, включали 100 пкмоль 3'-О-пропаргил-dATP, 100 пкмоль 3'-О-пропаргил-dCTP, 100 пкмоль 3'-О-пропаргил-dGTP и 100 пкмоль 3'-О-пропаргил-dTTP. Реакции, анализирующие отношение природных dNTP к 3'-О-пропаргил-dNTP 100:1, включали 10 пкмоль 3'-О-пропаргил-dATP, 10 пкмоль 3'-О-пропаргил-dCTP, 10 пкмоль 3'-О-пропаргил-dGTP и 10 пкмоль 3'-О-пропаргил-dTTP.
Проводили термоциклирование в реакциях полимеразного удлинения путем инкубирования при 95°С в течение 2 минут с последующими 45 циклами при 95°С в течение 15 секунд, при 55°С в течение 25 секунд и при 65°С в течение 35 секунд. После реакции полимеразного удлинения 5'-азидо-метил-модифицированные олигонуклеотиды химически конъюгировали с фрагментами нуклеиновой кислоты, терминированными 3'-О-пропаргил-dN, с использованием реакций «клик-химии», как описано в Примере 6 и Примере 7. После конъюгирования, продукты конъюгации использовали в качестве матриц для амплификации с получением ампликонов, соответствующих конъюгированным продуктам, как описано в Примере 8. Анализ размеров фрагмента осуществляли на конъюгированных продуктах.
Анализ размеров фрагментов амплифицированных продуктов конъюгации, полученных из продуктов для трех разных условий молярных отношений, показал, что размер фрагмента зависел от отношения 3'-О-пропаргил-dNTP к природным dNTP. Анализ размеров фрагментов показал сдвиг распределения фрагментов по размеру в зависимости от молярных отношений dNTP к 3'-О-пропаргил-dNTP. При молярном отношении 2:1 большие популяции более коротких фрагментов выявлялись по сравнению с двумя другими условиями молярного отношения. При молярном отношении 10:1 присутствовала большая фракция более длинных фрагментов по сравнению с молярным отношением 2:1. При молярном отношении 100:1 основная популяция фрагментов включала более длинные фрагменты ДНК по сравнению с другими двумя молярными отношениями.
«Лестницы» фрагментов, полученные с помощью трех разных молярных отношений, использовали в качестве раздельных входных данных для создания библиотек NGS (Illumina) для секвенирования на секвенаторе MiSeq, как описано в Примере 9. Затем были получены прочтения последовательности, как описано в Примере 10, и данные последовательности из последовательности-мишени извлекали и анализировали, как описано в Примере 11.
Профили покрытия для трех библиотек, которые были получены с использованием трех разных молярных отношений dNTP к 3'-О-пропаргил-dNTP (молярные отношения, составляющие 2:1, 10:1 и 100:1) коррелировали с распределением размеров «лестницы» фрагментов ДНК, созданной с использованием соответствующих молярных отношений. Например, ожидалось, что молярное отношение dNTP к 3'-О-пропаргил-dNTP, составляющее 2:1, будет терминировать полимеразное удлинение с высокой частотой из-за относительно большого количества 3'-О-пропаргил-dNTP и, таким образом, приводить к получению «лестницы» фрагментов нуклеиновой кислоты, относительно более коротких по сравнению с фрагментами, полученными при более высоких отношениях dNTP к 3'-О-пропаргил-dNTP. Напротив, ожидалось, что молярное отношение 100:1 будет терминировать полимеразное удлинение с низкой частотой из-за относительно низкого количества 3'-О-пропаргил-dNTP и, таким образом, приводить к получению «лестницы» фрагментов нуклеиновой кислоты, относительно более длинных по сравнению с фрагментами, полученными при более низких отношениях dNTP к 3'-О-пропаргил-dNTP.
Данные, собранные в результате анализа размеров фрагментов продуктов «лестницы» ДНК, созданных с использованием трех разных молярных отношений, подтвердили указанные предсказания. В частности, данные указывают на то, что изменение молярного отношения dNTP к 3'-О-пропаргил-dNTP обеспечивает контроль размера фрагментов «лестницы» ДНК.
Более того, секвенирование продуктов «лестницы» ДНК, созданных с использованием трех разных молярных отношений, и анализ последовательности, полученной из «лестницы» продуктов, показал, что профиль покрытия последовательности коррелировал с молярным отношением dNTP к 3'-О-пропаргил-dNTP, используемым во время создания «лестницы» ДНК. В частности, данные указывали на то, что молярное отношение 2:1 обеспечивало большее покрытие последовательности рядом с сайтом связывания праймера секвенирования, и молярное отношение 100:1 обеспечивало большее покрытие на удалении от сайта связывания праймера секвенирования. Соответственно, технология обеспечивает возможность контролировать создание «лестницы» фрагментов ДНК для различных способов применения секвенирования. В частности, повышение степени покрытия на удалении от сайта связывания праймера секвенирования является полезным для областей применения секвенирования, связанных с секвенированием длинных последовательностей (например, содержащих более чем 100 пар оснований). Секвенирование с использованием множества библиотек для секвенирования, полученных при разных молярных отношениях, обеспечивает данные последовательности с высоким покрытием последовательностей, расположенных рядом, внутри или на удалении от сайта связывания праймера для секвенирования.
Пример 13 - мечение праймерами, содержащими индексную последовательность
В процессе разработки вариантов реализации технологии, предложенной в настоящей заявке, проводили эксперименты для оценки применения индексных или штрихкод-последовательностей для трекинга и конструирования последовательности исходной матрицы-мишени на основе последовательности, полученной в результате создания библиотеки, NGS и выравнивания. В первой группе экспериментов нуклеиновые кислоты-мишени копировали и метили с помощью полимеразных реакций удлинения с использованием мишень-специфичных праймеров, содержащих уникально идентифицируемую индексную последовательность. При использовании в настоящей заявке, указанные и подобные способы молекулярного штрихкодирования называются «реакциями копирования и мечения» или «реакциями копирования и ID-мечения».
В указанной схеме создавали праймер для полимеразного удлинения, который содержал две области (Фигура 7): 3' участок, содержащий специфичную по отношению к мишени праймирующую последовательность, и 5' участок, содержащий две разных универсальных последовательности (например, универсальную последовательность А и универсальную последовательность В), фланкирующих вырожденную последовательность (например, содержащую 8 п.о.). Олигонуклеотидные праймеры синтезировали согласно указанной схеме и использовали в реакциях полимеразного удлинения со вторым олигонуклеотидом, созданным для остановки полимеразного удлинения, и, таким образом, «копирования и мечения» только интересующей области-мишени:
праймер полимеразного удлинения Eg_e19_R_SOD_v03-01-bio (SEQ ID NO: 6)
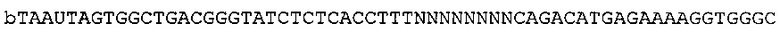
блокатор полимеразного удлинения Eg_e19_SOD_SC-200_v1 (SEQ ID NO: 7)
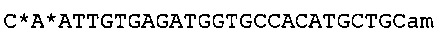
В последовательностях праймера полимеразного удлинения и блокатора полимеразного удлинения, используемых в реакции полимеразного удлинения во время процесса «копирования и мечения» (SEQ ID NO: 6 и 7 выше), «b» указывает на 5'-биотиновую модификацию, «U» указывает на дезоксиуридиновую модификацию, «*» указывает на фосфоротиоатную связь, и «am» указывает на 3'-амино-модификацию.
Реакции полимеразного удлинения проводили с использованием коммерчески доступного набора «мастер-миксы полимеразы для ПЦР высокой точности воспроизведения (набор для ПЦР KAPA HiFi HotStart, KAPA Biosystems) с получением реакционной смеси, содержащей 1 пкмоль праймера полимеразного удлинения (например, Eg_e19_R_SOD_v03-01-bio), 1 пкмоль блокатора полимеразного удлинения (например, Eg_e19_SOD_SC-200_vl) и 100 нг очищенной геномной ДНК, выделенной их клеточной линии аденокарциномы легкого человека/немелкоклеточного рака легкого (клеточная линия NCI-H1975, доступная из Американской коллекции типовых культур (АТСС) с кодом доступа CRL-5908) в реакционном объеме 25 мкл. Реакции полимеразного удлинения инкубировали при 95°С в течение 2 минут, при 98°С в течение 30 секунд, при 58°С в течение 90 секунд и при 65°С в течение 30 секунд. Полимеразу dNTP и KAPA HiFi добавляли сразу после завершения этапа инкубации при 58°С.
Продукты реакции полимеразного удлинения очищали с использованием очистки на основе гранул (Ampure ХР, Beckman Coulter) в соответствии с протоколом производителя для удаления праймеров полимеразного удлинения, блокаторов полимеразного удлинения и других компонентов реакции удлинения. Затем использовали очистку на основе захвата с помощью твердой фазы с использованием покрытых стрептавидином магнитных микросфер (Dynabeads, MyOne Streptavidin C1, Life Technologies) для выделения продуктов реакции полимеразного удлинения из геномной ДНК-матрицы. После выделения продуктов реакции полимеразного удлинения добавляли 2× связывающий и промывочный буфер (2 М NaCl, 20 мМ Tris-HCl, 2 мМ EDTA, рН 7,5) к элюенту, полученному в результате очистки гранул, в соотношении 1:1 (по объему) и инкубировали с гранулами, покрытыми стрептавидином, при температуре окружающей сред (комнатной температуре) в течение 15 минут. Гранулы отделяли от супернатанта с помощью магнита и супернатант удаляли. Затем гранулы промывали два раза с использованием связывающего и промывочного буфера и ресуспендировали в 25 мкл буфера ТЕ (10 мМ TrisHCl, 0,1 мМ EDTA, рН приблизительно 8). Гранулы инкубировали с раствором 0,1 М NaOH и 0,1 М NaCl в течение 1 минуты для удаления любых следовых количеств остаточной геномной ДНК. Затем гранулы отделяли от супернатанта с использованием магнита (супернатант удаляли), гранулы промывали два раза с помощью связывающего и промывочного буфера и ресуспендировали в 25 мкл буфера ТЕ (10 мМ Tris-HCl, 0,1 мМ EDTA, рН приблизительно 8).
В конечном итоге, для высвобождения связанного с гранулой продукта использовали систему расщепления (урацилгликозилат и эндонуклеазу VIII, Enzymatics) для отщепления связанного с гранулами продукта полимеразного удлинения в положении дезоксиуридиновой модификации, включенной в 5' конец продукта полимеразного удлинения в результате удлинения праймера полимеразного удлинени (см. SEQ ID NO: 6). Супернатант, содержащий продукт полимеразного удлинения, очищали с использованием системы Ampure ХР (Beckman Coulter) в соответствии с протоколом производителя и элюировали в 20 мкл буфера ТЕ.
Праймеры амплификации Uni_R_v2 и e19_F_v1 конструировали, синтезировали и использовали для амплификации очищенного продукта полимеразного удлинения для подтверждения образования продукта копирования и мечения, как схематично показано на Фигуре 8. Праймеры амплификации Uni_R_v2 и SC-240_COM_v1 использовали для подтверждения того, что блокатор полимеразного удлинения эффективно блокировал полимеразное удлинение после сайта связывания блокатора полимеразного удлинения с матрицей.
Uni_R_v2 (SEQ ID NO: 8)

e19_F_v1 (SEQ ID NO: 9)
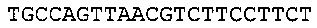
SC-240_COM_v1 (SEQ ID NO: 10)
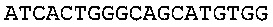
Проводили две реакции амплификации продукта полимеразного удлинения. Первая реакция включала праймеры Uni_R_v2 и e19_F-v1, которые амплифицируют как блокированные (с помощью блокатора полимеразного удлинения), так и не блокированные продукты полимеразного удлинения. Вторая реакция включала праймеры Uni_R_v2 и SC-240_COM_v1, которые амплифицировали только не блокированные продукты полимеразного удлинения. Два типа реакционных смесей получали с использованием коммерчески доступной смеси для амплификации (KAPA 2G HS, KAPA Biosystems) и 0,25 мкМ каждого праймера (как указано выше для двух реакций) в конечном реакционном объеме 25 мкл. Очищенный продукт полимеразного удлинения в объеме 5 мкл использовали в качестве матрицы для каждой реакции амплификации. Термоциклирование в реакциях амплификации осуществляли путем инкубирования реакционных смесей при 95°С в течение 5 минут с последующими 30 циклами при 98°С в течение 20 секунд, при 60°С в течение 30 секунд и при 72°С в течение 20 секунд. Продукты амплификации анализировали с помощью электрофореза в геле (например, с использованием системы Agilent Bioanalyzer 2100 и высокочувствительного ДНК-чипа) для определения распределения фрагментов по размеру.
Данные, полученные в результате анализа размера фрагментов, показали, что реакция амплификации, включающая праймеры Uni_R_v2 и e19_F_v1, приводила к получению продукта ожидаемого размера. Более того, данные также показали, что реакция амплификации, включающая праймеры Uni_R_v2 и SC_240_COM_v1, не приводила к получению выявляемого продукта, таким образом, указывая на то, что блокатор полимеразного удлинения эффективно останавливал полимеразную реакцию. Таким образом, технология обеспечивает точный контроль реакции копирования и мечения с получением продуктов только из интересующей области-мишени.
Пример 14 - мечение адаптерами, содержащими индексную последовательность
Дополнительно во второй группе экспериментов, проводимых в процессе разработки вариантов реализации, описанных в настоящей заявке, нуклеиновые кислоты-мишени копировали и затем метили путем лигирования с адаптером с использованием адаптеров, содержащих уникально идентифицируемую индексную последовательность. В указанной схеме молекулярного штрихкодирования на основе лигирования адаптера (см., например, Фигуру 9) ДНК адаптер конструировали с использованием двух олигонуклеотидов. Был создан первый олигонуклеотид, содержащий последовательность вырожденной последовательности (например, содержащий от 8 до 12 оснований), фланкированной как на 5' конце, таки и на 3' конце двумя разными универсальными последовательностями (например, универсальной последовательностью А и универсальной последовательностью В; см. Фигуру 9). Был создан второй олигонуклеотид, содержащий универсальную последовательность С (например, на 5' конце) и последовательность (например, на 3' конце), которая комплементарна универсальной последовательности В и которая имеет дополнительный Т в 3'-концевом положении. Для получения ДНК-адаптера два олигонуклеотида смешивали в равных молярных количествах, инкубировали при 95°С в течение 5 минут, и затем медленно охлаждали до температуры окружающей среды (комнатной температуры) для достижения эффективной гибридизации комплементарных частей двух олигонуклеотидов (например, универсальной последовательности В и ее комплементарной последовательности). Лигирование указанных адаптеров с ДНК-мишенью обеспечивает уникальное 'ID-мечение' каждой отдельной молекулы ДНК-мишени (например, каждого отдельного ПЦР-ампликона), например, в реакции, содержащей молярный избыток уникальный ID-меченной последовательности адаптеров по сравнению с количеством отдельных молекул-мишеней.
Проводили эксперименты для анализа вариантов реализации технологии согласно настоящему изобретению с использованием следующих олигонуклеотидов:
ST-adN10-phos-v1 (SEQ ID NO: 11)
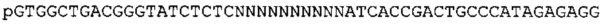
ST-ad-T-v1 (SEQ ID NO: 12)
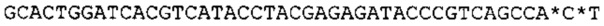
В последовательностях двух олигонуклеотидов, используемых для образования адаптера (SEQ ID NO: 11 и 12 выше), «р» указывает на 5' фосфатную модификацию, «N» указывает на положение вырожденного основания (например, положение может соответствовать А, С, G или Т), и «*» указывает на фосфоротиоатную связь.
В первом этапе осуществляли реакцию амплификации для амплификации участка размером 158 п.о. в экзоне 18 (с фланкирующей интронной последовательностью) человеческого гена EGFR с использованием следующих праймеров:
E_e18_f_v1p (SEQ ID NO: 13)
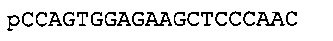
E_e18_r_v1p (SEQ ID NO: 14)
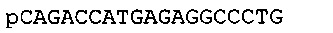
В последовательностях двух праймеров EGFR (SEQ ID NO: 13 и 14 выше) «р» указывает на 5'-фосфатную модификацию. Реакционные смеси получали с использованием коммерчески доступного набора «мастер-микси для ПЦР (набор для ПЦР KAPA 2G HotStart PCR, KAPA Biosystems), 10 пкмоль каждого из праймеров EGFR (SEQ ID NO: 13 и 14) и 10 нг очищенной геномной ДНК, выделенной из клеточной линии аденокарциномы легкого человека/немелкоклеточного рака легкого (клеточная линия NCI-H1975, доступная из АТСС под доступом CRL-5908) в реакционном объеме 25 мкл. Термоциклирование реакционных смесей осуществляли путем инкубирования при 95°С в течение 2 минут с последующими 23 циклами при 98°С в течение 20 секунд, при 63°С в течение 30 секунд и при 68°С в течение 20 секунд. После амплификации 1 мкл реакционной смеси использовали непосредственно для анализа размера фрагмента ДНК с использованием гель-электрофореза (например, Agilent 2100 Bioanalyzer и высокочувствительного аналитического ДНК-чипа). Данные, полученные в результате анализа фрагментов, указывали на то, что амплификация приводила к получению продукта, имеющего ожидаемый размер 158 п.о.
Затем продукт амплификации очищали для удаления не включенных праймеров и компонентов реакции амплификации с использованием способа очистки на основе гранул (Ampure ХР, Beckman Coulter) в соответствии с протоколом производителя.
После очистки адаптер, содержащий индексную последовательность (например, как описано выше), лигировали с ампликоном. Ампликон, полученный в результате реакции амплификации, описанной выше, содержал 5' фосфат (например, в результате включения 5' фосфат модифицированных праймеров) и 3'-dA-липкий конец (например, в результате действия ДНК-полимеразы, которая добавляет нематричную последовательность А на 3'-конец продуктов удлинения). Реакцию лигирования осуществляли с использованием коммерчески доступного набора для лигирования (Т4 DNA Ligase-Rapid, Enzymatics). В частности, реакционную смесь лигирования получали с использованием лигирующего буфера набора «Rapid», 25 пкмоль адаптеров и приблизительно 0,25 пкмоль ампликона в реакционном объеме 50 мкл.
После реакции лигирования полученную смесь инкубировали при 25°С в течение 10 минут и сразу очищали два раза с использованием очистки на основе гранул (Ampure ХР, Beckman Coulter) с последующим осуществлением протокола изготовителя, за исключением того, что объем вводимого образца на объем раствора гранул был иземен и составлял 1:1 вместо 1:1,8.
Очищенный лигированный продукт использовали в качестве матрицы для обогащающей амплификации с ограниченным количеством циклов (например, 8 циклами) для амплификации лигированного продукта (Фигура 10). Реакция амплификации включала праймеры, созданные для амплификации лигированного продукта, содержащего часть 'ID-метки' (например, 10 вырожденных оснований) и имеющего ожидаемую длину, составляющую 249 п.о.:
PCR1 (SEQ ID NO: 15)
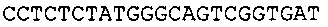
ST-PCR1-R-v1 (SEQ ID NO: 16)
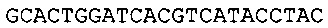
Амплификацию осуществляли с использованием коммерчески доступного набора «мастер-микс», содержащего полимеразу для ПЦР с высокой точностью воспроизведения (набор для ПЦР KAPA HiFi HotStart, KAPA Biosystems), для получения реакционной смеси, содержащей 0,25 мкМ каждого праймера и очищенный лигированный с адаптером продукт в качестве матрицы в реакционном объеме 25 мкл. Термоциклирование реакционных смесей для амплификации проводили путем инкубирования при 95°С в течение 5 минут с последующими 8 циклами при 98°С в течение 20 секунд, при 60°С в течение 30 секунд и при 72°С в течение 20 секунд. После амплификации 1 мкл реакционной смеси использовали непосредственно для анализа размера фрагмента с помощью электрофореза в геле (Agilent 2100 Bioanalyzer и высоко чувствительный чип для анализа ДНК). Данные, полученные в результате анализа фрагмента, указывали, что амплификация приводила к получению ампликона ожидаемого размера из лигорованного с адаптером продукта (например, ампликону размером 249 п.о., содержащего часть, соответствующую ампликону EGFR размером 158 п.о., полученному выше и лигированному с адаптером).
Пример 15 - циркуляризации нуклеиновой кислоты-мишени
Во процессе разработки вариантов реализации технологии, предложенной в настоящей заявке, проводили эксперименты для оценки молекулярного способа на основе внутримолекулярного лигирования (циркуляризации) нуклеиновой кислоты-мишени для расположения различных участков нуклеиновой кислоты-мишени в конкретном порядке. Способ включал циркуляризацию нуклеиновой кислоты-мишени, в результате которой известная последовательность (например, универсальная праймирующая последовательность) помещается рядом с неизвестной последовательностью (например, интересующим участком, например, искомым с помощью секвенирования) в конкретной ориентации (Фигура 11).
В указанных экспериментах реакции циркуляризации осуществляли с использованием коммерчески доступного набора оцДНК лигазы (CircLigase II, Epicentre-Illumina) в соответствии с протоколом производителя. В экспериментах анализировали синтетические исходные матрицы, которые представляли собой олигонуклеотиды («ультрамеры») длиной 100, 150 и 200 оснований:
Ультрамер-200 п.о. (SEQ ID NO: 17)

Ультрамер-150 п.о. (SEQ ID NO: 18)

Ультрамер-100 п.о. (SEQ ID NO: 19)

В последовательностях ультрамеров (SEQ ID NO: 17, 18 и 19, выше) «р» указывает на 5'-фосфатную модификацию.
После реакции циркуляризации продукты обрабатывали экзонуклеазой I и III (NEB) в течение 30 минут при 37°С для удаления не циркуляризованной матрицы. После экзонуклеазной обработки экзонуклеазы инактивировали путем инкубирования при 80°С в течение 10 минут. Для подтверждения циркуляризаци матриц создавали праймеры для ампификации специфичных по отношению к кольцевым матрицам продуктов амплификации (Фигура 12):
e19_F_v1 (SEQ ID NO: 20)
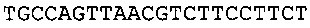
e19_circ_v1 (SEQ ID NO: 21)
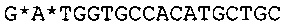
В последовательностях праймеров кольцевой матрицы (SEQ ID NOs: 20 и 21 выше) «*» обозначает фосфоротиоатную связь.
Амплификацию реакционных смесей проводили с использованием системы Taq-Gold (Abbott Molecular), 0,2 мкМ каждого праймера и с использованием одного из трех продуктов реакции разного размера в качестве матрицы в реакционном объеме 25 мкл. Проводили термоциклирвание реакционных смесей путем инкубирования при 95°С в течение 5 минут с последующими 38 циклами при 98°С в течение 20 секунд, при 60°С в течение 30 секунд и при 68°С в течение 30 секунд. После амплификации 10 мкл реакционной смеси использовали непосредственно для анализа размера фрагмента ДНК с помощью электрофореза в геле с использованием готовых 2% агарозных гелей (2% агарозный гель E-Gel EX, Life Technologies). Полученные данные показывают, что амплификация приводила к получению продукта из кольцевых матриц ожидаемого размера, что, таким образом, подтверждает образование кольцевых нуклеиновых кислот из трех тестируемых ультрамеров. Более того, отсутствие специфичных в отношении кольцевых матриц продуктов в отрицательных контролях, содержащих линейные матрицы, указывает на то, что праймеры приводили к получению специфичных в отношении кольцевых матриц продуктов.
Все публикации и патенты, перечисленные выше, полностью включены в настоящую заявку посредством ссылки во всех отношениях. Различные модификации и варианты описанных композиций, способов и способов применения технологии в пределах объема и сущности технологии согласно описанному изобретению, будут очевидны специалистам в данной области техники. Несмотря на то что технология была описана со ссылкой на конкретные примеры вариантов реализации, необходимо понимать, что заявленное изобретение не ограничивается указанными конкретными вариантами реализации. Предполагается, что различные модификации описанных способов реализации изобретения, которые очевидны специалистам в данной области техники, находятся в пределах объема следующей далее формулы изобретения.
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ABBOTT MOLECULAR INC.
KIM, Dae Hyun
<120> БИБЛИОТЕКИ ДЛЯ СЕКВЕНИРОВАНИЯ НОВОГО ПОКОЛЕНИЯ
<130> ABBTT-33341/WO-1/ORD
<140> PCT/US2014/051739
<141> 2014-08-19
<150> US 61/867,224
<151> 2013-08-19
<160> 50
<170> PatentIn version 3.5
<210> 1
<211> 177
<212> ДНК
<213> HOMO SAPIENS
<400> 1
ttattataag gcctgctgaa aatgactgaa tataaacttg tggtagttgg agctggtggc 60
gtaggcaaga gtgccttgac gatacagcta attcagaatc attttgtgga cgaatatgat 120
ccaacaatag aggtaaatct tgttttaata tgcatattac tggtgcagga ccattct 177
<210> 2
<211> 48
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 2
taaucctctc tatgggcagt cggtgataga atggtcctgc accagtaa 48
<210> 3
<211> 32
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 3
tctgagtcgg agacacgcag ggatgagatg gt 32
<210> 4
<211> 23
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 4
cctctctatg ggcagtcggt gat 23
<210> 5
<211> 22
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 5
ccatctcatc cctgcgtgtc tc 22
<210> 6
<211> 61
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<220>
<221> misc_feature
<222> (33)..(40)
<223> n представляет собой a, c, g, t или u
<400> 6
taautagtgg ctgacgggta tctctcacct ttnnnnnnnn cagacatgag aaaaggtggg 60
c 61
<210> 7
<211> 27
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 7
caattgtgag atggtgccac atgctgc 27
<210> 8
<211> 21
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 8
agtggctgac gggtatctct c 21
<210> 9
<211> 22
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 9
tgccagttaa cgtcttcctt ct 22
<210> 10
<211> 19
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 10
atcactgggc agcatgtgg 19
<210> 11
<211> 53
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<220>
<221> misc_feature
<222> (21)..(30)
<223> n представляет собой a, c, g или t
<400> 11
gtggctgacg ggtatctctc nnnnnnnnnn atcaccgact gcccatagag agg 53
<210> 12
<211> 44
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 12
gcactggatc acgtcatacc tacgagagat acccgtcagc cact 44
<210> 13
<211> 20
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 13
ccagtggaga agctcccaac 20
<210> 14
<211> 19
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 14
cagaccatga gaggccctg 19
<210> 15
<211> 23
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 15
cctctctatg ggcagtcggt gat 23
<210> 16
<211> 23
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 16
gcactggatc acgtcatacc tac 23
<210> 17
<211> 200
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 17
gcagcatgtg gcaccatctc acaattgcca gttaacgtct tccttctctc tggtgagaaa 60
gttaaaattc ccgtcgctat caaggaatta agagaagcaa catctccgaa agccaacaag 120
gaaatcctcg atgtgagttt ctgctttgct gtgtgggggt ccatggctct gaacctcagg 180
cccacctttt ctcatgtctg 200
<210> 18
<211> 150
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 18
gcagcatgtg gcaccatctc acaattgcca gttaacgtct tccttctctc tatctccgaa 60
agccaacaag gaaatcctcg atgtgagttt ctgctttgct gtgtgggggt ccatggctct 120
gaacctcagg cccacctttt ctcatgtctg 150
<210> 19
<211> 100
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 19
gcagcatgtg gcaccatctc acaattgcca gttaacgtct tccttctctc tgatgtgagt 60
ttctgctttg cttcctcagg cccacctttt ctcatgtctg 100
<210> 20
<211> 22
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 20
tgccagttaa cgtcttcctt ct 22
<210> 21
<211> 18
<212> ДНК
<213> искусственная
<220>
<223> синтетическая
<400> 21
gatggtgcca catgctgc 18
<210> 22
<211> 169
<212> ДНК
<213> HOMO SAPIENS
<400> 22
gaactacttg gaggaccgtc gcttggtgca ccgcgacctg gcagccagga acgtactggt 60
gaaaacaccg cagcatgtca agatcacaga ttttgggctg gccaaactgc tgggtgcgga 120
agagaaagaa taccatgcag aaggaggcaa agtaaggagg tggctttag 169
<210> 23
<211> 120
<212> ДНК
<213> HOMO SAPIENS
<400> 23
gacctggcag ccaggaacgt actggtgaaa acaccgcagc atgtgcaaga tcacagattt 60
tgggctggcc aaactgctgg gtgcggaaga gaaagaatac catgcagaag gaggcaaagt 120
<210> 24
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<220>
<221> misc_feature
<222> (35)..(35)
<223> n представляет собой a, c, g или t
<400> 24
gcagcatgtc aagatcacag attttgggct ggccn 35
<210> 25
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 25
gcagcatgtc aagatcacag attttgggct ggcca 35
<210> 26
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<220>
<221> misc_feature
<222> (35)..(35)
<223> n представляет собой a, c, g или t
<400> 26
cagcatgtca agatcacaga ttttgggctg gccan 35
<210> 27
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<220>
<221> misc_feature
<222> (1)..(4)
<223> n представляет собой a, c, g или t
<400> 27
nnnncatgtc aagatcacag attttgggct ggcca 35
<210> 28
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 28
cagcatgtca agatcacaga ttttgggctg gccaa 35
<210> 29
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<220>
<221> misc_feature
<222> (35)..(35)
<223> n представляет собой a, c, g или t
<400> 29
agcatgtcaa gatcacagat tttgggctgg ccaan 35
<210> 30
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 30
agcatgtcaa gatcacagat tttgggctgg ccaaa 35
<210> 31
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<220>
<221> misc_feature
<222> (34)..(35)
<223> n представляет собой a, c, g или t
<400> 31
catgtcaaga tcacagattt tgggctggcc aaann 35
<210> 32
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 32
gcatgtcaag atcacagatt ttgggctggc caaac 35
<210> 33
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 33
catgtcaaga tcacagattt tgggctggcc aaact 35
<210> 34
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 34
atgtcaagat cacagatttt gggctggcca aactg 35
<210> 35
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 35
atgtcaagat cacagatttt ggctggccaa actgc 35
<210> 36
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 36
atgtcaagtc acagattttg ggctggccaa actgc 35
<210> 37
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 37
tgtcaagatc acagattttg ggctggccaa actgc 35
<210> 38
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<220>
<221> misc_feature
<222> (1)..(1)
<223> n представляет собой a, c, g или t
<400> 38
ngtcaagatc acagattttg ggctggccaa actgc 35
<210> 39
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<220>
<221> misc_feature
<222> (1)..(3)
<223> n представляет собой a, c, g или t
<400> 39
nnncaagatc acagattttg ggctggccaa actgc 35
<210> 40
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 40
tgtcagatca cagattttgg gctggccaaa ctgct 35
<210> 41
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 41
gtcaagatca cagattttgg gctggccaaa ctgct 35
<210> 42
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<220>
<221> misc_feature
<222> (1)..(1)
<223> n представляет собой a, c, g или t
<400> 42
ncaagatcac agattttggg ctggccaaac tgctg 35
<210> 43
<211> 35
<212> ДНК
<213> HOMO SAPIENS
<400> 43
tcaagatcac agattttggc tggccaaact gctgg 35
<210> 44
<211> 177
<212> ДНК
<213> HOMO SAPIENS
<400> 44
ttattataag gcctgctgaa aatgactgaa tataaacttg tggtagttgg agctggtggc 60
gtaggcaaga gtgccttgac gatacagcta attcagaatc attttgtgga cgaatatgat 120
ccaacaatag aggtaaatct tgttttaata tgcatattac tggtgcagga ccattct 177
<210> 45
<211> 31
<212> ДНК
<213> HOMO SAPIENS
<400> 45
acgggctcgt tggatgctag ctgatcgcga a 31
<210> 46
<211> 30
<212> ДНК
<213> HOMO SAPIENS
<400> 46
cggctcgctg gatgctagct gatcgcgaat 30
<210> 47
<211> 30
<212> ДНК
<213> HOMO SAPIENS
<400> 47
ggctcgctgg atgctggctg atcgcgaatg 30
<210> 48
<211> 30
<212> ДНК
<213> HOMO SAPIENS
<400> 48
gcacgctgga tgctagctga tcgcgaatgc 30
<210> 49
<211> 30
<212> ДНК
<213> HOMO SAPIENS
<400> 49
ctcgctggat gctagctgat cgagaatgca 30
<210> 50
<211> 34
<212> ДНК
<213> HOMO SAPIENS
<400> 50
acggctcgct ggatgctagc tgatcgcgaa tgca 34
| название | год | авторы | номер документа |
|---|---|---|---|
| НАБОР ДЛЯ ПОЛУЧЕНИЯ РЕАКЦИОННОЙ СМЕСИ ДЛЯ СИНТЕЗА 3′-O-ПРОПАРГИЛ-МОДИФИЦИРОВАННОЙ НУКЛЕИНОВОЙ КИСЛОТЫ | 2014 |
|
RU2688435C2 |
| СЕКВЕНИРОВАНИЕ ПОЛИНУКЛЕОТИДНЫХ БИБЛИОТЕК С ВЫСОКОЙ ПРОПУСКНОЙ СПОСОБНОСТЬЮ И АНАЛИЗ ТРАНСКРИПТОМОВ | 2018 |
|
RU2790291C2 |
| ПОЛНОГЕНОМНЫЕ БИБЛИОТЕКИ ОТДЕЛЬНЫХ КЛЕТОК ДЛЯ БИСУЛЬФИТНОГО СЕКВЕНИРОВАНИЯ | 2018 |
|
RU2770879C2 |
| ВЫСОКОПРОИЗВОДИТЕЛЬНОЕ СЕКВЕНИРОВАНИЕ ОДИНОЧНОЙ КЛЕТКИ СО СНИЖЕННОЙ ОШИБКОЙ АМПЛИФИКАЦИИ | 2019 |
|
RU2744175C1 |
| АНАЛИЗ МНОЖЕСТВА АНАЛИТОВ С ИСПОЛЬЗОВАНИЕМ ОДНОГО АНАЛИЗА | 2019 |
|
RU2824049C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ОПРЕДЕЛЕНИЯ ЛИГАНДОВ НА МАТРИЦАХ С ИСПОЛЬЗОВАНИЕМ ИНДЕКСОВ И ШТРИХКОДОВ | 2020 |
|
RU2825578C1 |
| ВЫСОКОПРОИЗВОДИТЕЛЬНЫЕ БИБЛИОТЕКИ ОДИНОЧНЫХ КЛЕТОК И СПОСОБЫ ПОЛУЧЕНИЯ И ИСПОЛЬЗОВАНИЯ | 2020 |
|
RU2838946C2 |
| СПОСОБЫ ВЫЯВЛЕНИЯ РАКА ЛЕГКОГО | 2017 |
|
RU2760913C2 |
| КРУПНОМАСШТАБНЫЕ МОНОКЛЕТОЧНЫЕ БИБЛИОТЕКИ ТРАНСКРИПТОМОВ И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2019 |
|
RU2773318C2 |
| СПОСОБЫ И СРЕДСТВА ПОЛУЧЕНИЯ БИБЛИОТЕКИ ДЛЯ СЕКВЕНИРОВАНИЯ | 2019 |
|
RU2815513C2 |








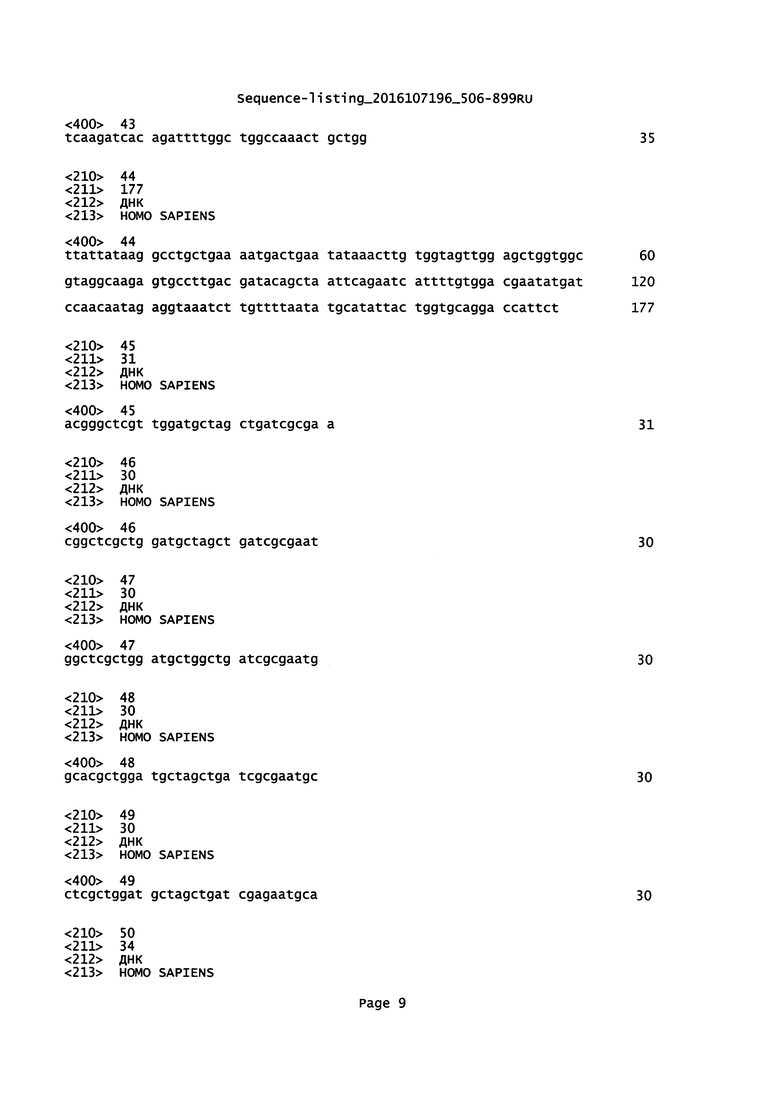

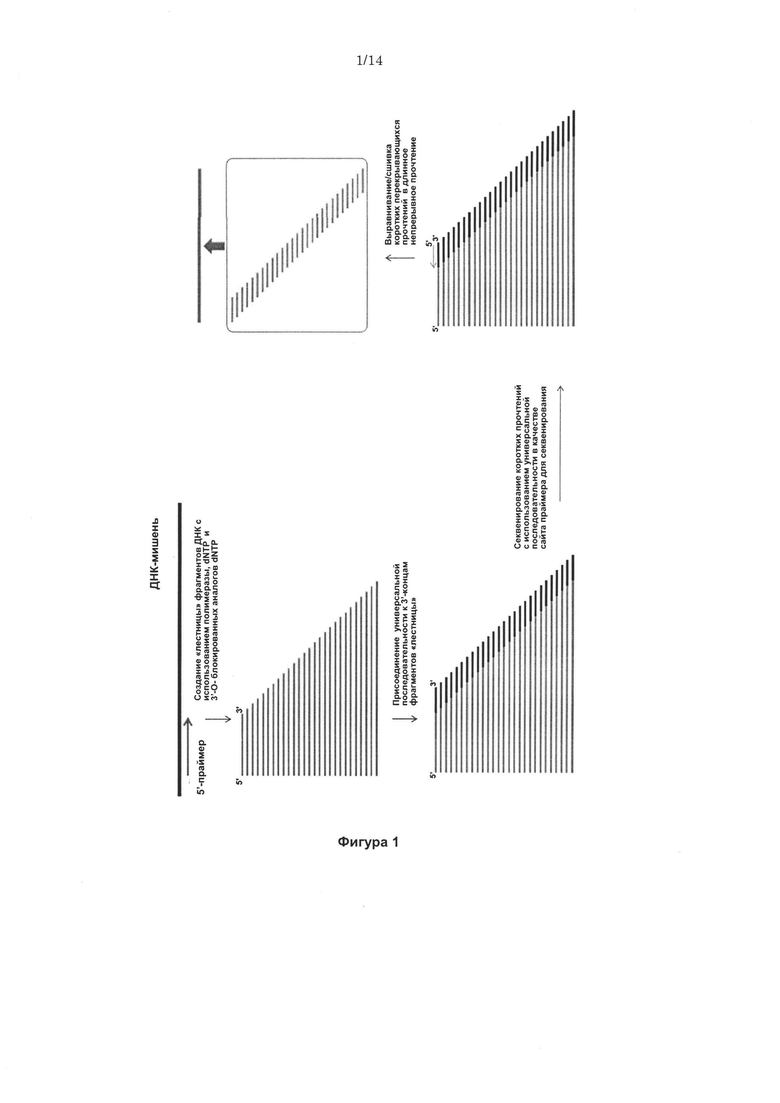
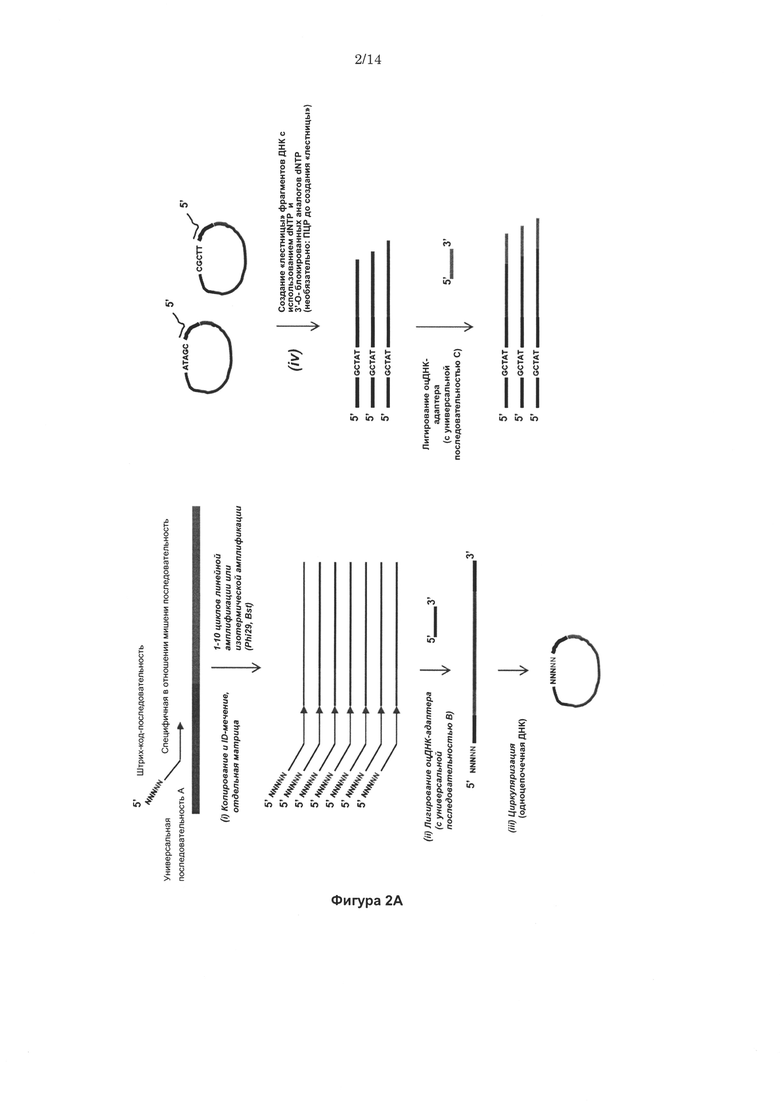
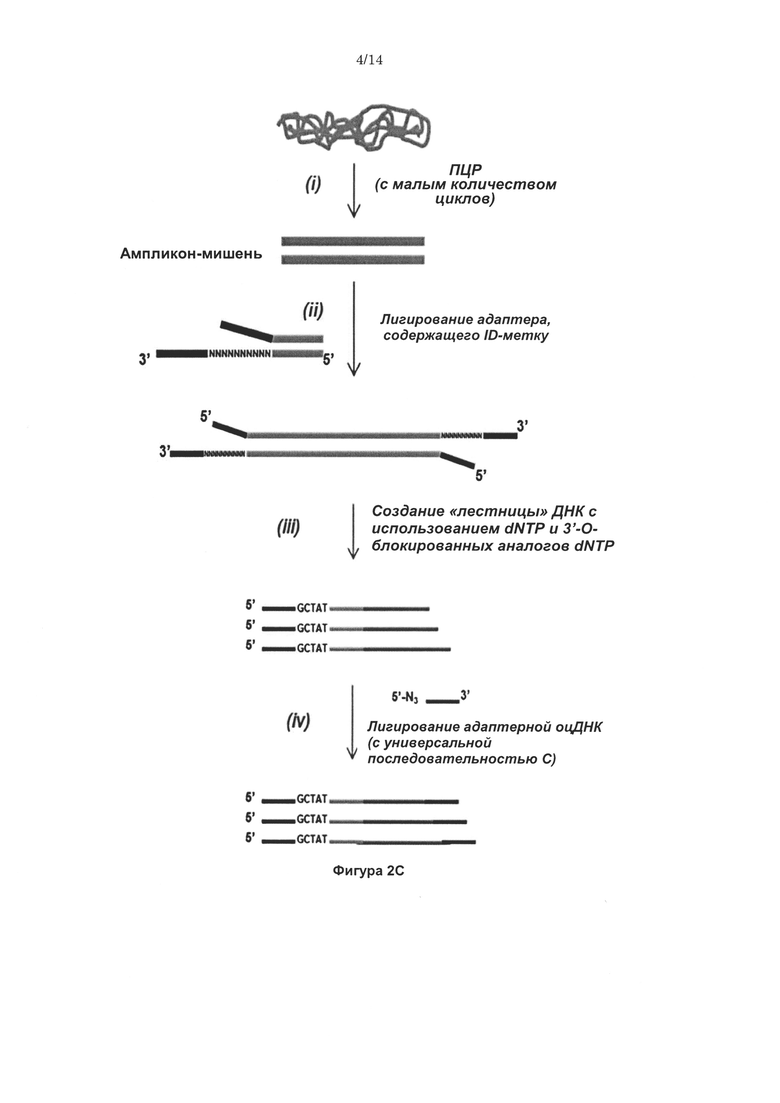

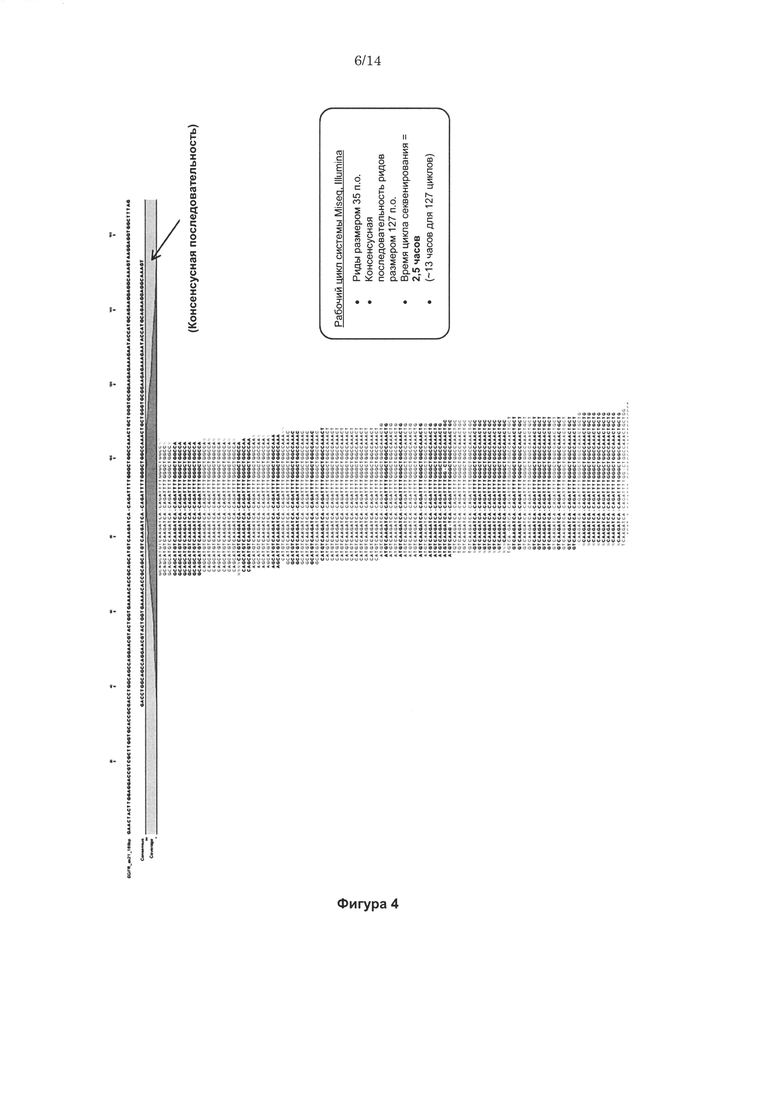
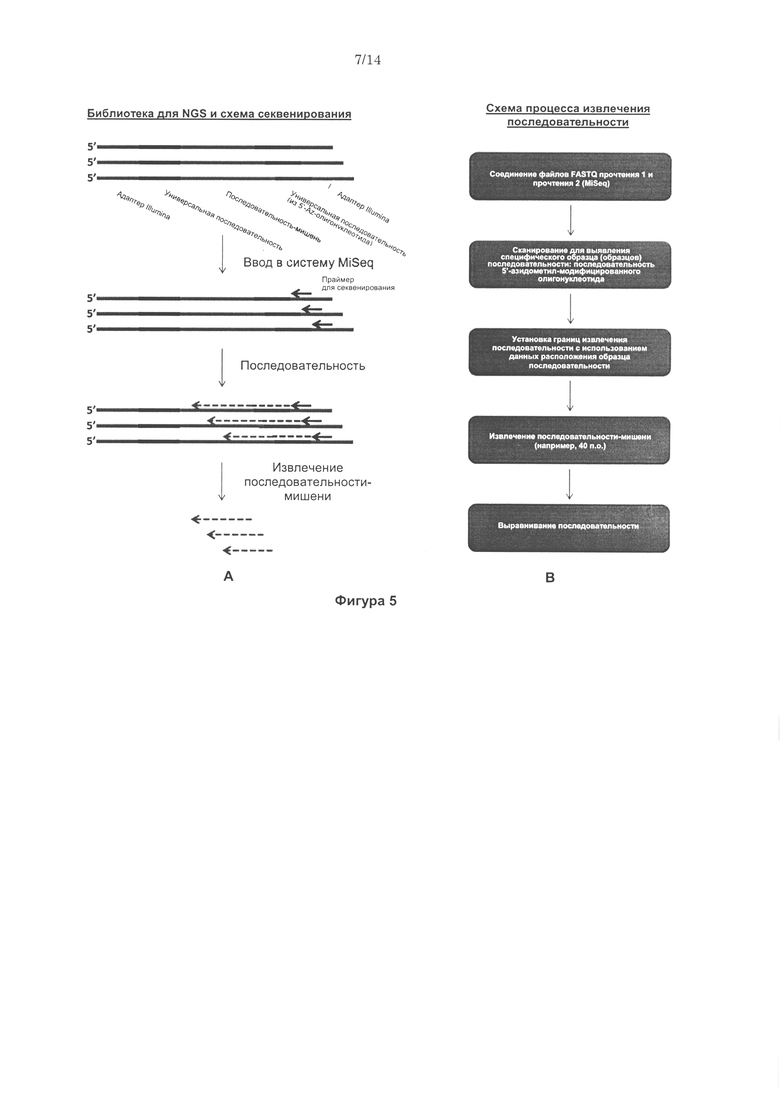

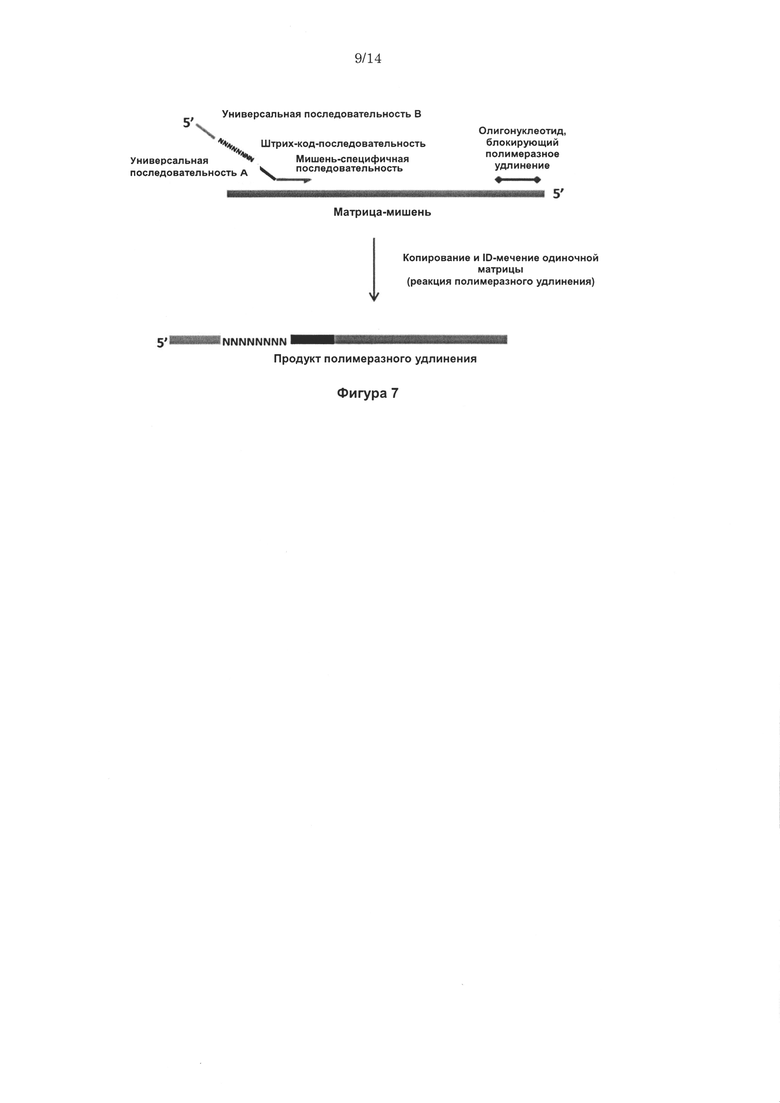

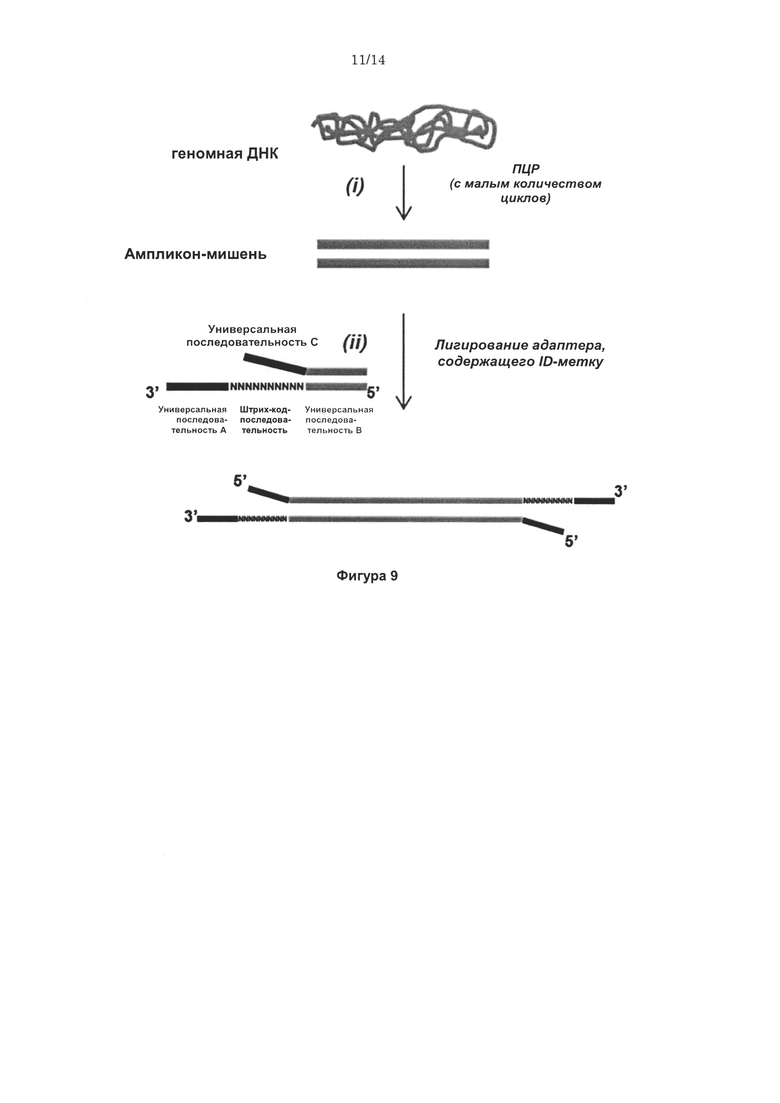
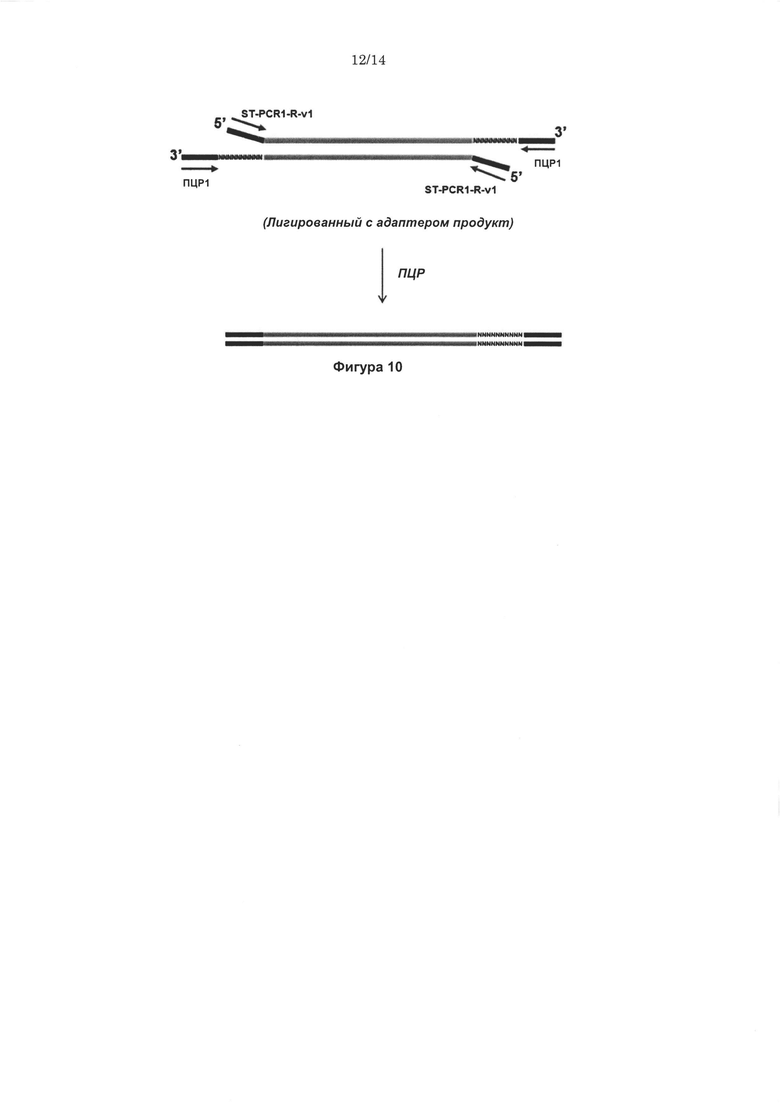

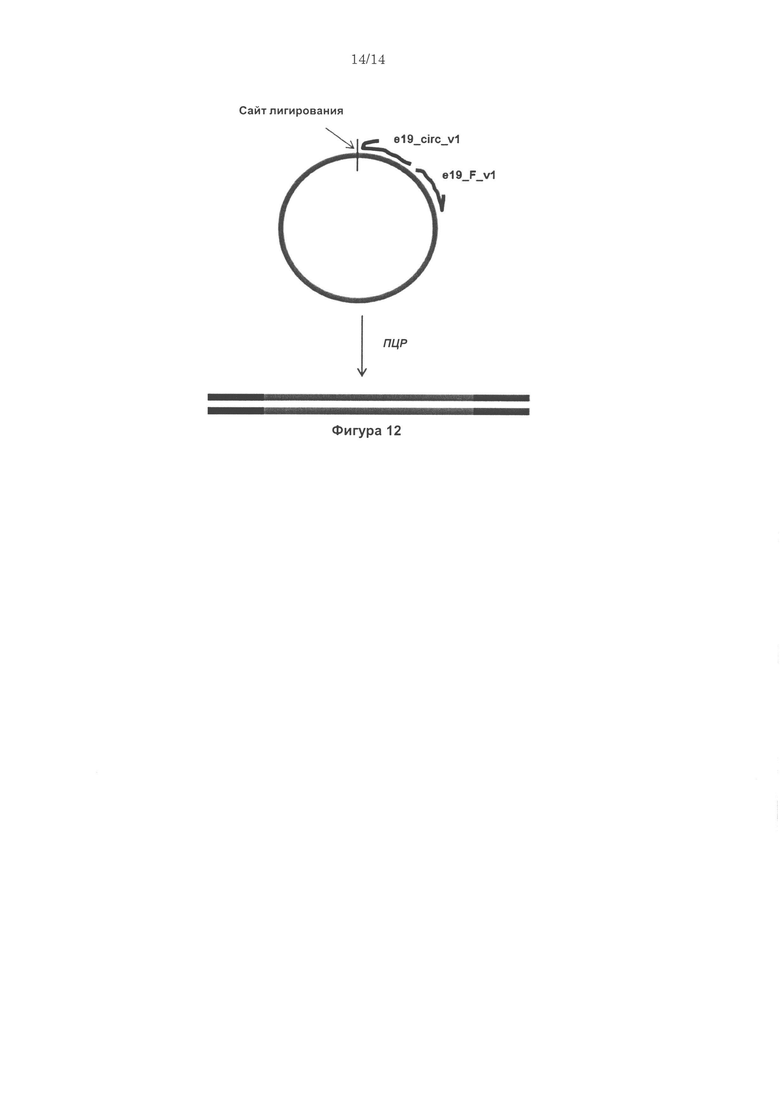
Изобретение относится к области биотехнологии и молекулярной биологии. Предложен способ определения нуклеотидной последовательности-мишени, включающий создание библиотеки для секвенирования нового поколения, что включает амплификацию нуклеотидной последовательности-мишени с использованием праймера, содержащего специфичную в отношении мишени последовательность и последовательность A, с получением ампликона, где указанный ампликон может быть одноцепочечным или двуцепочечным, при этом указанная последовательность А представляет собой известную последовательность, которая является одинаковой среди множества праймеров, при этом со всеми праймерами, содержащими указанную последовательность А, можно проводить манипуляции и/или амплификацию, идентификацию, секвенирование или выделение одинаковым или сходным образом; лигирование первого адаптерного олигонуклеотида, содержащего последовательность B, с указанным ампликоном для образования адаптера-ампликона, при этом указанная последовательность В представляет собой известную последовательность, которая является одинаковой среди множества праймеров, при этом со всеми праймерами, содержащими указанную последовательность В, можно проводить манипуляции и/или амплификацию, идентификацию, секвенирование или выделение одинаковым или сходным образом; и создание библиотеки, представляющей собой «лестницу» фрагментов, содержащей множество фрагментов для применения в качестве библиотеки для секвенирования нового поколения; и секвенирование фрагмента указанной библиотеки, представляющей собой «лестницу» фрагментов, где нуклеотидная последовательность, определенная в результате секвенирования, содержит нуклеотидную субпоследовательность нуклеотидной последовательности-мишени. Благодаря повышенному качеству секвенирования с коротким временем его осуществления способ может использоваться в медицине и в промышленности. 10 з.п. ф-лы, 12 ил., 6 табл., 15 пр.
1. Способ определения нуклеотидной последовательности-мишени, включающий:
a) создание библиотеки для секвенирования нового поколения, что включает
1) амплификацию нуклеотидной последовательности-мишени с использованием праймера, содержащего специфичную в отношении мишени последовательность и последовательность A, с получением ампликона, где указанный ампликон может быть одноцепочечным или двуцепочечным, при этом указанная последовательность А представляет собой известную последовательность, которая является одинаковой среди множества праймеров, при этом со всеми праймерами, содержащими указанную последовательность А, можно проводить манипуляции и/или амплификацию, идентификацию, секвенирование или выделение одинаковым или сходным образом;
2) лигирование первого адаптерного олигонуклеотида, содержащего последовательность B, с указанным ампликоном для образования адаптера-ампликона, при этом указанная последовательность В представляет собой известную последовательность, которая является одинаковой среди множества праймеров, при этом со всеми праймерами, содержащими указанную последовательность В, можно проводить манипуляции и/или амплификацию, идентификацию, секвенирование или выделение одинаковым или сходным образом; и
3) создание библиотеки, представляющей собой «лестницу» фрагментов, содержащей множество фрагментов для применения в качестве библиотеки для секвенирования нового поколения; и
b) секвенирование фрагмента указанной библиотеки, представляющей собой «лестницу» фрагментов, где нуклеотидная последовательность, определенная в результате секвенирования, содержит нуклеотидную субпоследовательность нуклеотидной последовательности-мишени.
2. Способ по п. 1, отличающийся тем, что указанный праймер дополнительно содержит нуклеотидную последовательность-штрихкод, связанную с указанной нуклеиновой кислотой-мишенью, и указанный способ дополнительно включает определение нуклеотидной последовательности-штрихкода фрагмента указанной библиотеки, представляющей собой «лестницу» фрагментов.
3. Способ по п. 1, дополнительно включающий лигирование второго адаптерного олигонуклеотида, содержащего последовательность C, с 3′-концами указанных фрагментов библиотеки, представляющей собой «лестницу» фрагментов, для создания библиотеки для секвенирования нового поколения, при этом определение нуклеотидной последовательности фрагмента указанной библиотеки, представляющей собой «лестницу» фрагментов, включает использование олигонуклеотидного праймера, комплементарного указанной последовательности C, и при этом указанная последовательность С представляет собой известную последовательность, которая является одинаковой среди множества праймеров, таким образом, что со всеми праймерами, содержащими указанную последовательность С, можно проводить манипуляции и/или амплификацию, идентификацию, секвенирование или выделение одинаковым или сходным образом.
4. Способ по п. 2, отличающийся тем, что определение указанной нуклеотидной последовательности-штрихкода фрагмента указанной библиотеки, представляющей собой «лестницу» фрагментов, включает использование олигонуклеотидного праймера, комплементарного последовательности B.
5. Способ по п. 1, отличающийся тем, что указанная нуклеотидная последовательность фрагмента библиотеки, представляющей собой «лестницу» фрагментов, содержит от 15 до 1000 нуклеотидов.
6. Способ по п. 2, включающий объединение указанной нуклеотидной последовательности-штрихкода с источником указанной нуклеотидной последовательности-мишени.
7. Способ по п. 2, дополнительно включающий группировку нуклеотидных последовательностей фрагментов указанной библиотеки, представляющей собой «лестницу» фрагментов, имеющих одинаковую нуклеотидную последовательность-штрихкод.
8. Способ по п. 1, дополнительно включающий сборку множества нуклеотидных последовательностей фрагментов указанной библиотеки, представляющей собой «лестницу» фрагментов, с обеспечением консенсусной последовательности.
9. Способ по п. 8, дополнительно включающий картирование указанной консенсусной последовательности на референсной последовательности.
10. Способ по п. 8, отличающийся тем, что указанная консенсусная последовательность сохраняет информацию о фазировании и/или сцеплении указанной нуклеиновой кислоты-мишени.
11. Способ по п. 1, отличающийся тем, что указанная библиотека, представляющая собой «лестницу» фрагментов, содержит нуклеотидную последовательность указанной нуклеиновой кислоты-мишени и
1) 5′-конец нуклеотидной субпоследовательности m находится на нуклеотиде xm указанной нуклеотидной последовательности-мишени и 3′-конец указанной нуклеотидной субпоследовательности находится на нуклеотиде ym указанной нуклеотидной последовательности-мишени;
2) 5′-конец нуклеотидной субпоследовательности (m + 1) находится на нуклеотиде xm+1 указанной нуклеотидной последовательности-мишени и 3′-конец указанной нуклеотидной субпоследовательности находится на нуклеотиде ym+1 указанной нуклеотидной последовательности-мишени;
3) m варьируется от 1 до n;
4) xm = xm+1; и
5) (ym+1 – ym) < 20.
| US 2009047680 A1, 19.02.2009 | |||
| US 2012165202 A1, 28.06.2012 | |||
| RU 2000130113 A, 27.12.2002. |
Авторы
Даты
2019-08-22—Публикация
2014-08-19—Подача