Область техники, к которой относится изобретение
Изобретение относится к области биотехнологии и генной инженерии растений, а именно к способу определения геномной локализации и числа копий Трансгенной-ДНК (Т-ДНК) в геноме трансформированных растений.
Предшествующий уровень техники
Место геномной локализации и число копий Т-ДНК в геноме трансформированных растений являются важнейшими характеристиками для дальнейшего использования трансгенных растений. Игнорирование данных характеристик может привести к нестабильному наследованию и экспрессии трансгена, фенотипическим отклонениям, не связанным с функционированием Т-ДНК, а также к генетическому расщеплению и утрате трансгена в последующих поколениях. Кроме этого, быстрое и точное определение зиготности введенных последовательностей, а также структуры занимаемых ими локусов позволяет сократить количество поколений, необходимых при получении линий со стабильной экспрессий от первоначальных трансформантов. В случае растений это наиболее актуально, так как наступление генеративного периода у них может занимать от нескольких месяцев до десятков лет. Современные методы трансформации не позволяют контролировать число копий встраиваемых генов, их ориентацию и место локализации в геноме. Ввиду этого в геноме трансформированных растений может наблюдаться наличие повторяющихся, усеченных или перестроенных копий вводимой последовательности, а также возможно нарушение регионов, кодирующих нативные гены. Молекулярный анализ интродуцированного гена и области его интеграции позволяет проводить обнаружение и изучение значительной доли нежелательных эффектов. Последние могут быть опосредованы, например, встраиванием гена в непредпочтительное для этого место в геноме (нативная рамка считывания или область низкой экспрессии). Кроме того, при использовании метода трансформации с помощью Agrobacterium tumefaciens часто наблюдается интеграция двух и более копий трансгена в один локус, что может приводить к замолканию экспрессии трансгена.
Для определения места трансгена в геноме и числа копий были разработаны различные методы. В настоящий момент для молекулярного анализа интродуцированных аллелей широко используются такие методы как Саузерн-блоттинг (Southern Е.М., et. al., 1975), количественная ПЦР (qPCR) ((Ingham, D.J. et. al., 2001)), термическая асимметричная ПЦР с чересстрочной разверткой (TAIL-PCR) (Liu Y.G., et. al., 2007), а также цифровая капельная ПЦР (ddPCR) (Collier, R. et. al., 2017) Саузерн-блоттинг является одним из наиболее точных методов определения количества копий введенных в геном аллелей. В сравнении с методами, основанными на ПЦР, недостатками Саузерн-блоттинга являются трудоемкость и сложность, а зачастую и невозможность автоматизации. Кроме этого, применение этого метода для определения копийности Т-ДНК у растений с большим геномом является практически невозможным ((Southern Е.М., et. al., 1975)).
Метод qPCR при анализе трансформированных растений предполагает относительную оценку количества исходной матрицы (в данном случае - встроенной в геном последовательности), на основе флуоресценции интеркалирующего красителя или репортерного ДНК-зонда. С помощью qPCR возможно определить количество копий интродуцированных аллелей, однако отклонения в эффективности амплификации, а также экспоненциальный характер увеличения количества исходного продукта являются факторами, ограничивающими точность, особенно при небольших различиях в копийности. Кроме того, поскольку qPCR позволяет проводить количественную оценку последовательности введенного гена, для подтверждения зиготности необходимо так или иначе прибегать к анализу расщепления в потомстве (Ingham, D.J. et. al., 2001).
Термическая асимметричная ПЦР с чересстрочной разверткой может быть использована как для определения числа копий, так и для локализации встроенных копий в геноме. Данный метод предполагает использование праймеров, специфичных для вставки, используемых совместно с произвольными вырожденными праймерами. Полученные ПЦР-продукты подлежат клонированию, секвенированию, и как правило содержат часть последовательности внесенного гена совместно с фланкирующим его участком генома. Главным ограничением TAIL-PCR является отсутствие амплификации при анализе некоторой части инсерций интродуцированного гена, что зачастую приводит к заниженным результатам при подсчете количества его копий. Также вставка Т-ДНК в регион с повторяющимися последовательностями может привести к артефактам (Liu Y.G., et. al., 2007).
Цифровая капельная ПЦР также позволяет анализировать количество копий введенных в геном аллелей. Как и при qPCR, в ddPCR используется интеркалирующий краситель или специфичные ДНК-зонды, однако сама полимеразная цепная реакция протекает независимо в множестве микрообъемов (капель), на которые делится реакционная смесь. Анализ единичных молекул ДНК, реализуемый при ddPCR, позволяет достичь точности, сравнимой с Саузерн-блоттингом, однако данный метод не позволяет определить локализацию введенных генов (Collier, R. et. al., 2017).
Отдельная группа методов связана с полногеномным секвенированием трансгенных растений методами NGS с последующим биоинформатическим анализом мест вставки Т-ДНК. Хотя этот метод является очень информативным, он имеет ряд недостатков, а именно: (1) необходимость полногеномного секвенирования с высоким покрытием (>10х), что экономически невыгодно при работе с растениями с крупным геномом и при анализе большого числа растений; (2) зависимость метода от первоначальной сборки генома.
Таким образом, на сегодня отсутствует универсальный подход, позволяющий определять места вставки Т-ДНК у разных видов растений с большим и малым размером генома, не прибегая к затратному полногеномному секвенированию.
Сущность изобретения
Задачей изобретения, является разработка нового способа определение места и числа копий Т-ДНК в геноме растений, лишенного ограничений предшествующего уровня техники и создание маркеров для генотипирования трансформированных растений с помощью Cas9-целевого нанопорового секвенирования.
Для решения данной задачи предлагается способ, в основе которого лежит целевое нанопоровое секвенирование с помощью рибонуклеопротеиновых комплексов Cas9/гРНК.
На первом этапе происходит выделение высокомолекулярной фракции ДНК (HMW DNA) из индивидуальных трансгенных растений ЦТАБ-методом (Kirov I. et. al., 2021). Количество и качество экстрагированной ДНК оценивается на спектрофотометрах: NanoDrop One UV-Vis Spectrophotometer (Thermo Scientific, USA) и Quantus Fluorometer (Promega, USA). Для дальнейших исследований подходит только чистая ДНК (А260/А280 ~ 1,8 и А260/А230 ~ 2,0 по данным NanoDrop) практически без различий в концентрациях, полученных с помощью приборов Nanodrop и Quantus.
На следующем этапе ДНК индивидуальных растений объединяется в равных количествах, чтобы в результате получить не менее 10 мкг. ДНК в 60 мкл. свободной от нуклеаз воды. Обогащение объединенной пробы длинными фрагментами осуществляется при помощи Short Read Eliminator Kit (Circulomics, США) по методике рекомендованной производителем.
Далее происходит биоинформатический подбор гРНК, с помощью программы FlashFry (McKenna A. et. al., 2018) специфичные для области Т-ДНК и синтез гРНК in vitro на молекуле двуцепочечной ДНК-матрицы содержащей последовательность Т7-промотора и последовательности, полученной в результате объединения двух олигонуклеотидов, специфичного и универсального комплементарных бордерам Т-ДНК. Для сборки рибонуклеопротеиновых (РНП) комплексов используется 50 нг гРНК 20 пмоль Cas9 нуклеазы (Биолабмикс, Новосибирск, Россия).
Подготовка ДНК библиотеки для Cas9-целевого нанопорового секвенирования на приборе MinION (Oxford Nanopore Technologies, Великобритания) осуществляется по протоколам, описанным в статье Kirov I. et. al., 2021. Сначала анализируемая ДНК дефосфолирируется ферментом Quick CIP (New England Biolabs, Великобритания), затем дефосфолирированная ДНК расщепляется с помощью рибонуклеопротеидных комплексов Cas9/гРНК. Затем на концы ДНК, свободные от рибонуклеопротеидных комплексов Cas9/гРНК лигируются белковые адаптеры для секвенирования ((Oxford Nanopore Technologies, Великобритания) с помощью набора SQK-LSK109 (Oxford Nanopore Technologies, Великобритания). Секвенирование происходит на приборе MinION (Oxford Nanopore Technologies, Великобритания), с проточными ячейками R9.4.1 или R10.3.
Для идентификация мест вставки Т-ДНК у анализируемых растений используется разработанный нами подход NanoCasTE (https://github.com/Kirovez/NanoCasTE). Для анализа используются инсерции, подтвержденные 2 и более ридами.
Для генотипирования индивидуальных растений, из изучаемой объединенной ДНК пробы, с помощью программы Primer3, подбираются два набора специфичных праймеров для каждой инсерции: одна пара праймеров, фланкирующих место инсерции, для детекции вариантов без инсерции и вторая пара праймеров с одним праймером (R) на участок 35S промотера (универсальный праймер для детекции всех инсерций) и (F) праймером на фланкирующую область. Специфичность праймеров оценивается с помощью программы Primer-BLAST.
Таким образом, описанный выше способ определения геномной локализации и числа копий Т-ДНК в геноме трансформированных растений с помощью Cas9-целевого нанопорового секвенирования, позволяет определять места вставки Т-ДНК у разных видов растений с большим и малым размером генома, не прибегая к затратному полногеномному секвенированию и трудоемкому Саузерн-блоттингу.
Краткое описание чертежей
Фигура 1. Схема вектора pCambia несущая кодирующую последовательность для GAG-eGFP для трансформации.
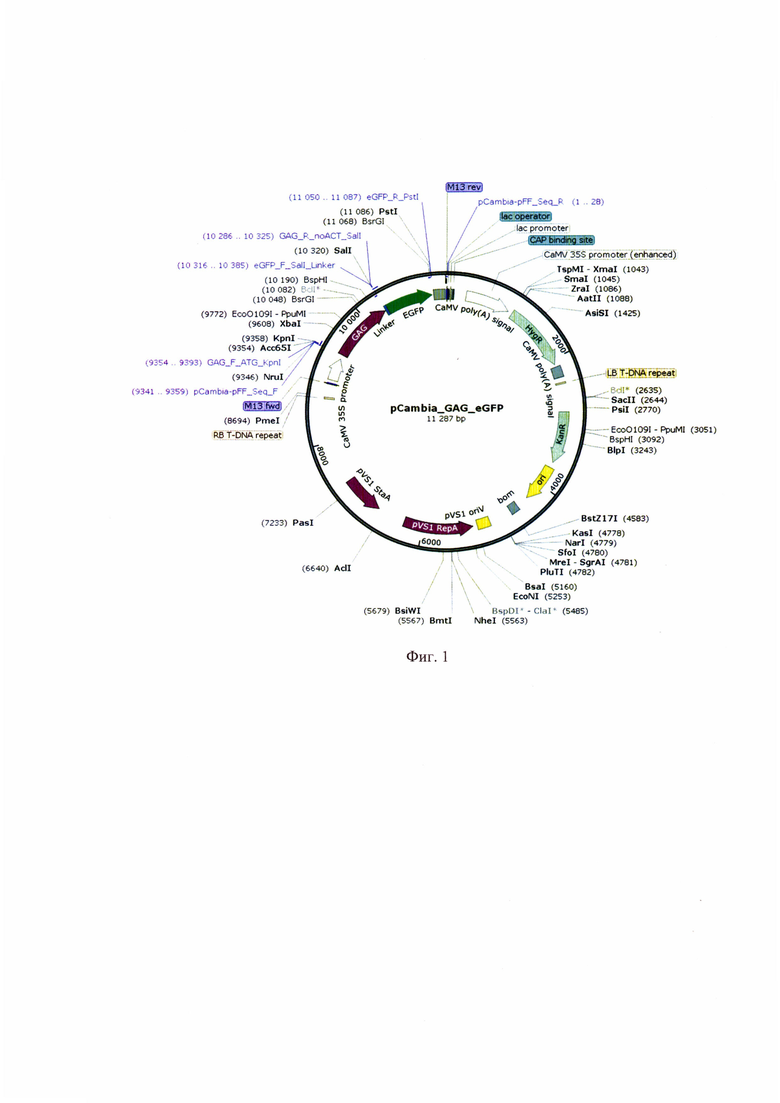
Фигура 2. Схема вектора pCambia несущая кодирующую последовательность для eGFP-GAG для трансформации.
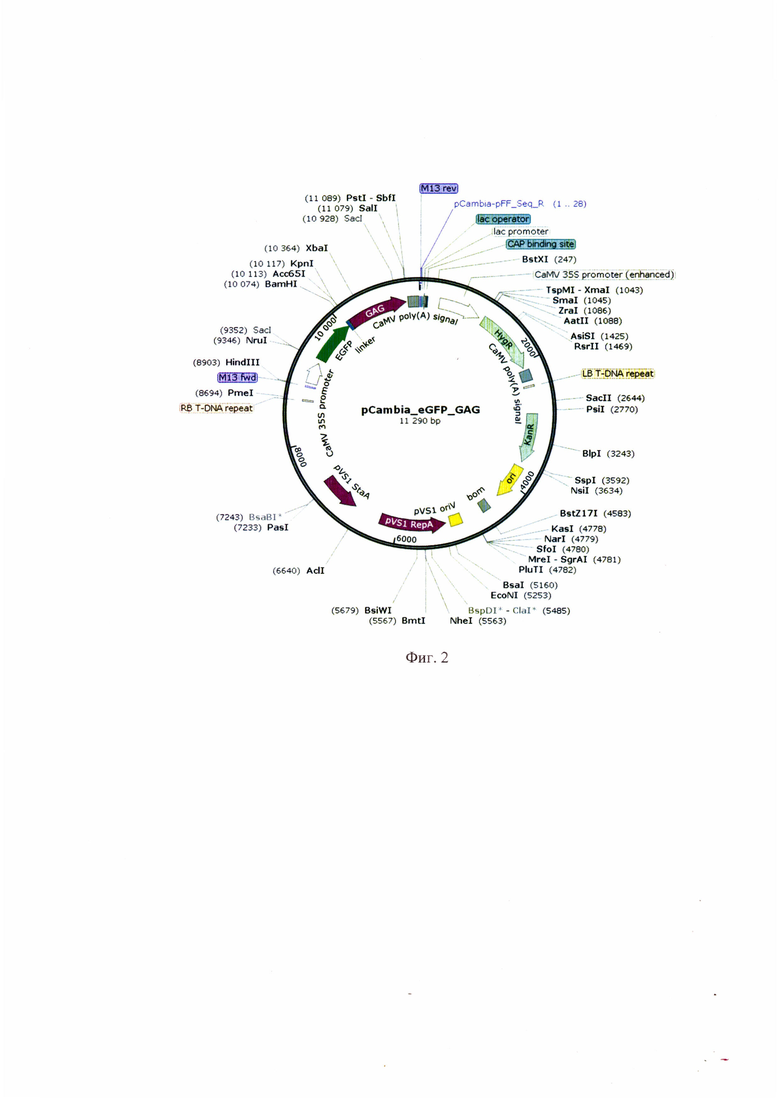
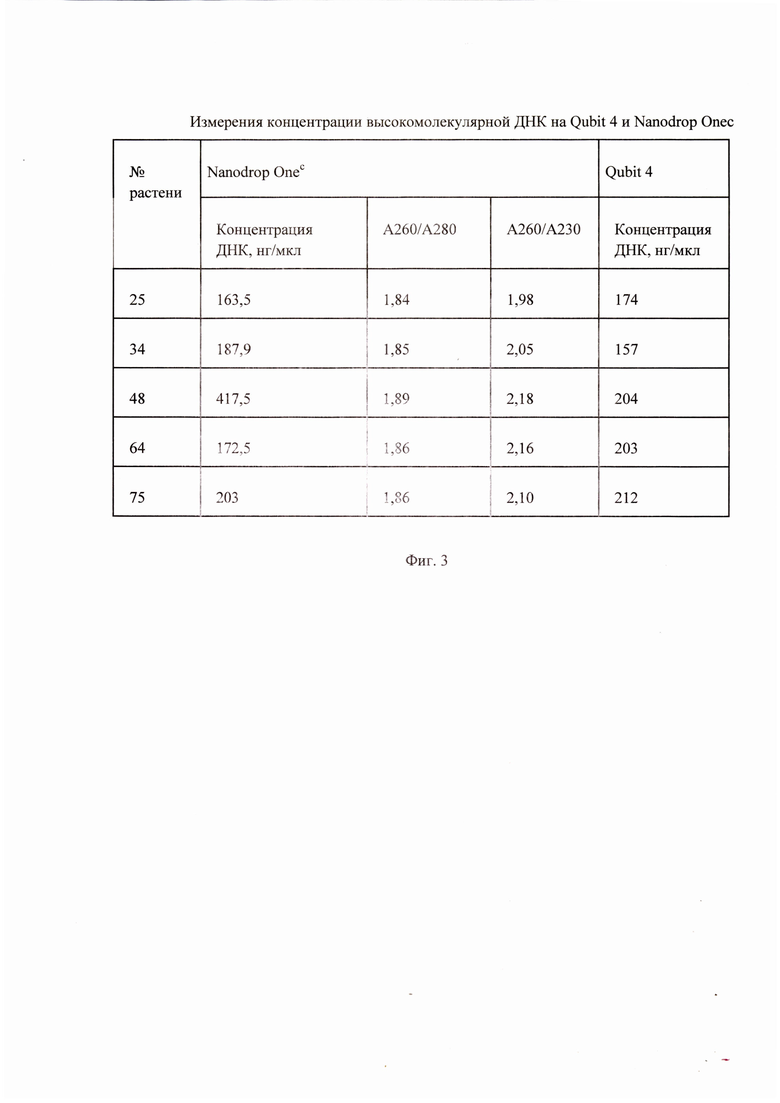
Фигура 3. Измерения концентрации высокомолекулярной ДНК на Qubit 4 и Nanodrop Onec.
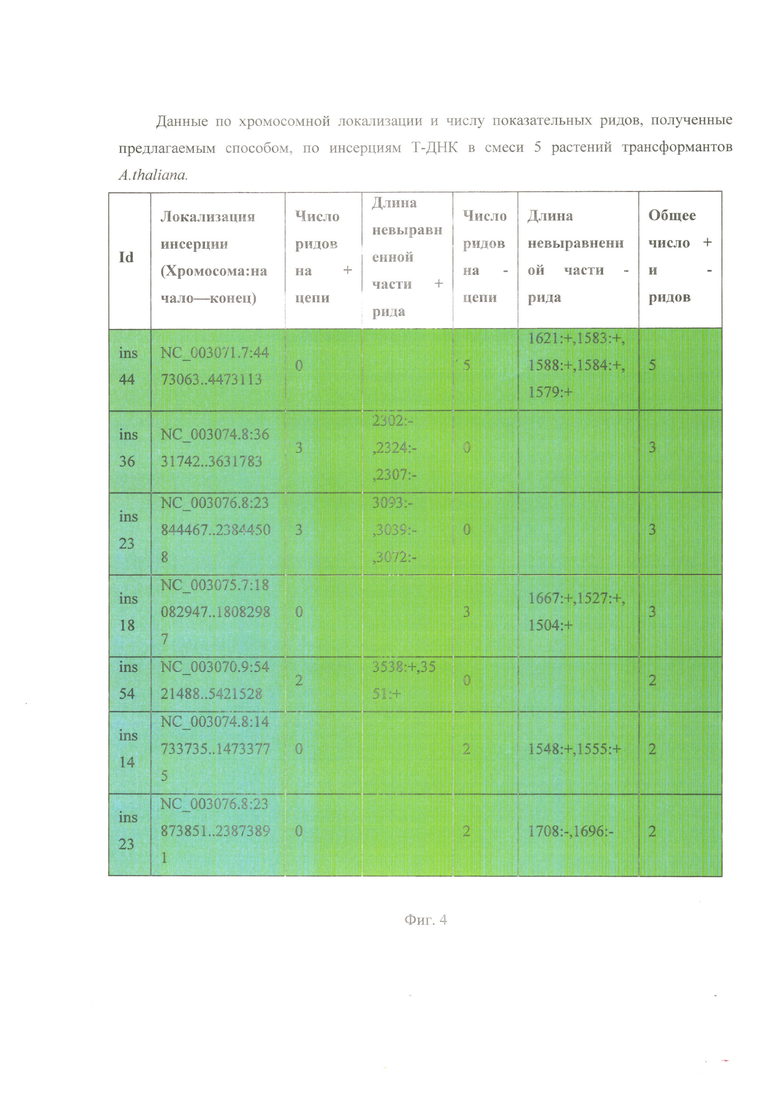
Фигура 4. Данные по хромосомной локализации и числу показательных ридов, полученные предлагаемым способом, по инсерциям Т-ДНК в смеси 5 растений трансформантов A. thaliana.
Фигура 5. Пример детектированных инсерций Т-ДНК у A. thaliana.
Пример 1
Определения геномной локализации и числа копий Т-ДНК у пула растений A. thaliana трансформированных вектором pCambia с целевым геном GAG, с помощью Cas9-целевого нанопорового секвенирования.
В качестве объекта были выбраны растения - трансформанты Arabidopsis thaliana поколения Т1, несущие вставку Т-ДНК с целевым геном GAG. Растения были получены путем трансформации методом floral-dip с последующим отбором трансформантов на селективной среде, содержащей гигромицин (Kirov I. et. al., 2021). Растения были трансформированы одним из двух векторов pCambia, отличающихся расположением рамки считывания для белка eGFP относительно GAG: GAG-eGFP (Фиг. 1) или eGFP-GAG (Фиг. 2).
Семена, полученные от трансформированных агробактериальным методом растений Arabidopsis thaliana экотипа Columbia, были высеяны в горшки (6 см×6 см) с грунтом. Растения выращивались в световой камере в течение месяца при температуре 20±1°С. и условиях длинного дня (16 часов света/8 часов темноты). Для выделения высокомолекулярной ДНК отбирались здоровые развитые листовые пластинки растений. Выделение высокомолекулярной ДНК проводили в вытяжном шкафу ЦТАБ-методом. Измерение концентрации высокомолекулярной ДНК проводили при помощи спектрофотометра Nanodrop Onec и флуориметра Qubit 4 (Фиг. 3).
Выделенная высокомолекулярная ДНК пяти образцов была в равных пропорциях смешана в 60 мкл воды, свободной от нуклеаз. Обогащение длинными фрагментами осуществляли при помощи Short Read Eliminator Kit (Circulomics, США).
Для секвенирования были разработаны три гРНК, специфичные для области Т-ДНК.
Гидовые РНК (гРНК) были получены посредством транскрипции in vitro на молекуле двуцепочечной ДНК-матрице, содержащей последовательность Т7-промотора и полученной в результате объединения двух олигонуклеотидов, специфичного TgagF1-2, TgagR1, и универсального CRISPR R:
TgagF1
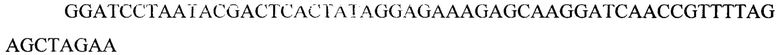
TgagF2
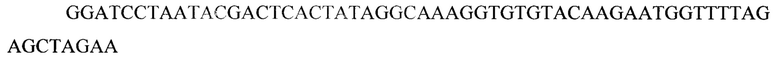
TgagR1
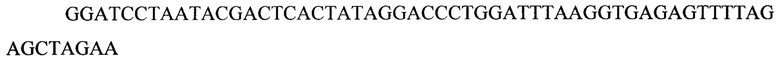
CRISPR R
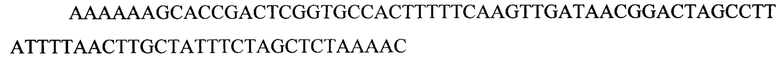
T7 F 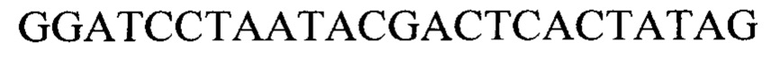
T7 R 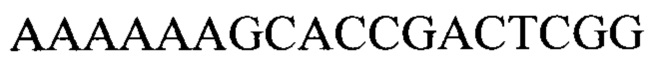
Для транскрипции был использован набор T7-transcription для высокоэффективного синтеза РНК in vitro (Биолабмикс, Новосибирск, Россия) в соответствии с рекомендациями производителя. Анализ концентрации и качества полученных гРНК осуществляли при помощи спектрофотометра Nanodrop OneC (Thermo Scientific, США), Qubit 4 (Thermo Scientific, США) и разделении в 2% агарозном геле. Для сборки рибонуклеопротеиновых (РНП) комплексов было использовано 50 нг каждой гРНК. Гидовые РНК были смешаны в эквимолярном соотношении 1:1 в 11 мкл воды, свободной от РНКаз. Для удаления вторичных структур смесь гРНК была подвергнута денатурации при 95°С в течение 3 минут, далее помещена на лед. Сборка РНП-комплексов проводилась добавлением 1 мкл Cas9 нуклеазы и 3 мкл реакционного буфера (Биолабмикс, Новосибирск, Россия) к 11 мкл смеси гРНК с последующей инкубацией при комнатной температуре в течение 30 минут. По истечении времени пробирка, содержащая РНП-комплексы, была помещена на лед. Геномная ДНК предварительно была подвергнута реакции дефосфорилирования с помощью фермента Quick CIP (New England Biolabs, Великобритания) с последующей инкубацией при 37°С в течение 30 минут. Инактивацию фермента проводили при 80°С в течение 2 минут. Разрезание ДНК РНП-комплексами проводили путем объединения 40 мкл дефосфорилированной ДНК, 15 мкл смеси РНП-комплексов, 1,5 мкл дАТФ (10 мМ) и 1 мкл полимеразы Encyclo (Evrogen, Moscow, Russia). Смесь инкубировали при 37°С в течение 30 минут, далее - 10 минут при 72°С. Далее проводили приготовление библиотек с помощью набора SQK-LSK109 (Oxford Nanopore Technologies, Великобритания).
Полученную ДНК библиотеку секвенировали приборе MinION с ячейкой R9.4. Время секвенирования составило около 24 часов и было собрано более 78395 ридов с N50 ~1600 п.н. Для детекции Т-ДНК инсерций был использован разработанный нами подход NanoCasTE (https://github.com/Kirovez/NanoCasTE). Для определения числа инсерция была проведена детекция инсерций, подтвержденная 2 и более ридами. После достижения числа 56,000 ридов число инсерций не изменялось и достигло 7. Таким образом данным методом было успешно идентифицированы инсерции Т-ДНК в смеси образцов из 5 растений. Инсерции локализовались на 5 хромосомах A. thaliana. Список инсерций, идентифицированных в результате анализа, приведен на Фиг. 4. Детектированные инсерции были подтверждены 2 и более ридами (Фиг. 5).
Для генотипирования индивидуальных растений были подобраны два набора специфичных праймеров для каждой инсерции: одна пара праймеров, фланкирующих место инсерции, для детекции вариантов без инсерции и вторая пара праймеров с одним праймером (R) на участок 35S промотера (универсальный праймер для детекции всех инсерций) и F праймером на фланкирующую область. Праймеры подбирали с помощью программы Primer3 и проверяли специфичность с помощью Primer-BLAST. Праймеры из первой группы амплифицировали фрагменты до 700 п.н. Список праймеров приведен ниже:
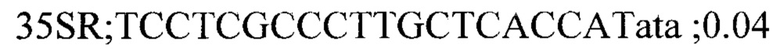
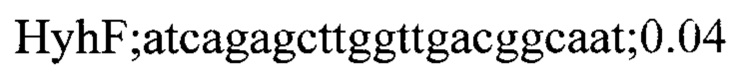
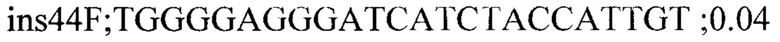
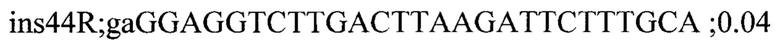
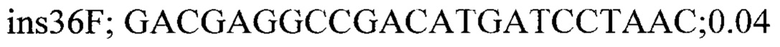
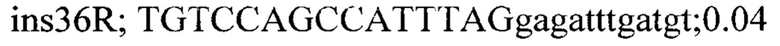
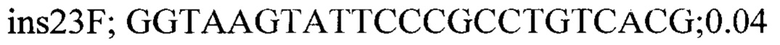
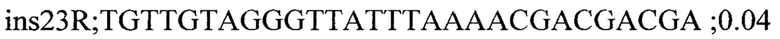
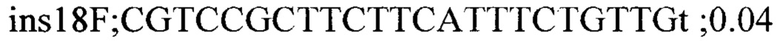
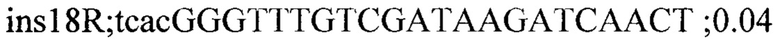
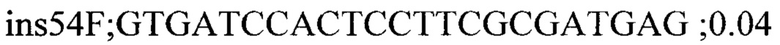

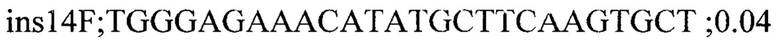
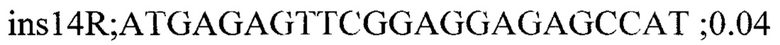
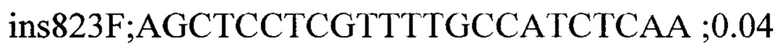
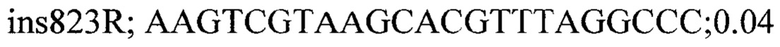
На следующем этапе проводилась ПЦР с комбинацией праймеров и ДНК индивидуальных растений. В результате все 7 инсерций были валидированы и получены данные по хромосомной локализации и числу локусов Т-ДНК у каждого из анализированных растений.
Финансирование работ по созданию настоящего изобретения проводилось из средств Соглашения №075-15-2019-1667 от «31» октября 2019 г. о предоставлении из федерального бюджета грантов в форме субсидий в соответствии с пунктом 4 статьи 78.1 Бюджетного кодекса Российской Федерации на осуществление государственной поддержки создания и развития центра геномных исследований мирового уровня «Курчатовский геномный центр» в рамках реализации федерального проекта «Развитие научной и научно-производственной кооперации» национального проекта «Наука».
Список литературы
Southern Е.М., Edwin Mellor. Detection of specific sequences among DNA fragments separated by gel electrophoresis // J mol biol. - 1975. - T. 98. - №.3. - C. 503-517.
Ingham, D.J., Beer, S., Money, S., & Hansen, G. Quantitative real-time PCR assay for determining transgene copy number in transformed plants // Biotechniques. - 2001. - T. 31. - №.1. - C. 132-140.
Liu Y.G., Chen Y. High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences // Biotechniques. - 2007. - T. 43. - №.5. - C. 649-656.
Collier, R., Dasgupta, K., Xing, Y.P., Hernandez, В.Т., Shao, M., Rohozinski, D., … & Thilmony / Accurate measurement of transgene copy number in crop plants using droplet digital PCR // The Plant Journal. - 2017. - T. 90. - №.5. - C. 1014-1025.
Ilya Kirov, Pavel Merkulov, Sofya Gvaramiya, Roman Komakhin, Murad Omarov, Maxim Dudnikov, Alina Kocheshkova, Alexander Soloviev, Gennady Karlov, Mikhail Divashuk (2021). Illuminating the transposon insertion landscape in plants using Cas9-targeted Nanopore sequencing and a novel pipeline. bioRxiv - 2021.
McKenna A, Shendure J. FlashFry: a fast and flexible tool for large-scale CRISPR target design. BMC biology. 2018 Dec; 16(1):1-6.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ОДНОВРЕМЕННОГО НАНОПОРОВОГО СЕКВЕНИРОВАНИЯ ПОЛНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ЗНАЧИМЫХ ГЕНОВ ТВЕРДОЙ ПШЕНИЦЫ | 2021 |
|
RU2785924C1 |
| ВЫСОКОПРОИЗВОДИТЕЛЬНЫЙ АНАЛИЗ ТРАНСГЕННЫХ ГРАНИЦ | 2012 |
|
RU2603265C2 |
| РНК-НАПРАВЛЯЕМАЯ ИНЖЕНЕРИЯ ГЕНОМА ЧЕЛОВЕКА | 2013 |
|
RU2699523C2 |
| СПОСОБ ОБНАРУЖЕНИЯ ТРАНСФОРМАНТА СОИ рDAB9582.814.19.1 | 2012 |
|
RU2622079C2 |
| ОБОГАЩЕНИЕ АКТИВИРУЕМОЙ ФЛУОРЕСЦЕНЦИЕЙ СОРТИРОВКИ КЛЕТОК (FACS) ДЛЯ СОЗДАНИЯ РАСТЕНИЙ | 2013 |
|
RU2679510C2 |
| МОЛЕКУЛА РНК-ПРОВОДНИКА ДЛЯ ГЕНОМНОГО РЕДАКТИРОВАНИЯ ПРОТОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ОДНОДОЛЬНЫХ ЗЕРНОВЫХ С ПРИМЕНЕНИЕМ СИСТЕМЫ CRISPR/CAS9 | 2020 |
|
RU2762831C1 |
| РНК-НАПРАВЛЯЕМАЯ ИНЖЕНЕРИЯ ГЕНОМА ЧЕЛОВЕКА | 2013 |
|
RU2766685C2 |
| ЛИНИИ ТРАНСГЕННОЙ СОИ, ГЕНЕТИЧЕСКОЕ СОБЫТИЕ 8264.42.32.1, УСТОЙЧИВОЕ К ГЕРБИЦИДАМ С ПАКЕТИРОВАННЫМИ ГЕНАМИ НА ЕГО ОСНОВЕ, И ИХ ДЕТЕКТИРОВАНИЕ | 2012 |
|
RU2636021C2 |
| СКОНСТРУИРОВАННАЯ СПОСОБАМИ ИНЖЕНЕРИИ ПЛАТФОРМА ДЛЯ ВСТРАИВАНИЯ ТРАНСГЕНА (ETIP) ДЛЯ НАЦЕЛИВАНИЯ ГЕНОВ И СТЭКИНГА ПРИЗНАКОВ | 2013 |
|
RU2666916C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ОПОСРЕДОВАННОЙ НУКЛЕАЗОЙ НАПРАВЛЕННОЙ ИНТЕГРАЦИИ ТРАНСГЕНОВ | 2013 |
|
RU2650819C2 |

Настоящее изобретение относится к области биотехнологии. Представлен способ определения геномной локализации и числа копий Т-ДНК в геноме трансформированных растений с помощью Cas9-целевого нанопорового секвенирования. Способ включает в себя следующие этапы: выделение высокомолекулярной фракции ДНК индивидуальных трансгенных растений (HMW DNA), пулирование ДНК индивидуальных трансгенных растений; подбор гРНК, специфичных для области Т-ДНК, и синтез гРНК in vitro на молекуле двуцепочечной ДНК-матрицы, сборка рибонуклеопротеидных комплексов Cas9/гРНК, Cas9-целевое нанопоровое секвенирование, идентификация мест вставки Т-ДНК у пула растений при помощи подхода NanoCasTE, подбор специфичных праймеров для каждой инсерции Т-ДНК на участки, фланкирующие место вставки, генотипирование индивидуальных растений. Изобретение позволяет получить новый способ определения места и числа копий Т-ДНК в геноме растений и создать маркеры для генотипирования трансформированных растений с помощью Cas9-целевого нанопорового секвенирования. 5 ил., 1 пр.
Способ определения геномной локализации и числа копий Т-ДНК в геноме трансформированных растений с помощью Cas9-целевого нанопорового секвенирования, включающий в себя:
выделение высокомолекулярной фракции ДНК индивидуальных трансгенных растений (HMW DNA),
объединение образцов ДНК индивидуальных трансгенных растений;
подбор гРНК, специфичных для области Т-ДНК, и синтез гРНК in vitro на молекуле двуцепочечной ДНК-матрицы,
сборку рибонуклеопротеиновых комплексов Cas9/гРНК,
Cas9-целевое нанопоровое секвенирование,
идентификацию мест вставки Т-ДНК у анализируемых растений при помощи подхода NanoCasTE,
подбор специфичных праймеров для каждой инсерции Т-ДНК на участки, фланкирующие место вставки,
генотипирование индивидуальных растений.
| SOUTHERN Е.М., EDWIN MELLOR, "Detection of specific sequences among DNA fragments separated by gel electrophoresis", J mol biol | |||
| Сплав для отливки колец для сальниковых набивок | 1922 |
|
SU1975A1 |
| - T | |||
| Дорожная спиртовая кухня | 1918 |
|
SU98A1 |
| Электрический фонарь - испытательный прибор | 1912 |
|
SU503A1 |
| MCKENNA A, SHENDURE J | |||
| "FlashFry: a fast and flexible tool for large-scale CRISPR target design", BMC biology | |||
| Способ получения цианистых соединений | 1924 |
|
SU2018A1 |
| LIU Y.G., CHEN Y | |||
| "High-efficiency thermal | |||
Авторы
Даты
2022-12-15—Публикация
2021-11-16—Подача