Область техники, к которой относится изобретение
Изобретение относится к области молекулярной биотехнологии и селекции растений, а именно к способу одновременного секвенирования нуклеотидных последовательностей панели генов, лежащих в основе важных для селекционного процесса признаков твердой пшеницы.
Предшествующий уровень техники
Не смотря на постоянное усовершенствование методов секвенирования, по-прежнему существует потребность в анализах представителей большинства видов растений, важных для сельскохозяйственной отрасли. Многие растения, которые были собраны, как эталонные геномы, зачастую выбирались, потому что являлись гомозиготными и, как правило, могут не представлять полный набор структурных вариаций, которые важны для селекции (Lopez-Girona Е. et al., 2020). Геномы даже близкородственных форм часто могут существенно отличаться, как по последовательности, так и по структуре от референтных.
Чтобы решить эту проблему, исследователи сосредоточились на классических способах секвенировании. Данные методы обеспечивают покрытие гена, но не дают возможность более точно идентифицировать распространенные геномные вариации, такие как однонуклеотидные полиморфизмы (SNP), которые присутствуют в геномах других сортов и линий. Потеря различных модификаций происходит при неточной сборке последовательности, что чаще обусловлено наличия повторов и мобильных элементов в составе аллелей, что является проблемой при использовании методов секвенирования с коротким чтением. Следовательно, потребовались новые методы выявления аллельных вариаций, которые могли бы быть применимы к широкому спектру культур (De Coster W et al., 2019).
В настоящее время наиболее оптимальным подходом в изучении последовательности генов стало использование технологий Oxford Nanopore Technologies. Однако, чтобы снизить затраты на секвенирование и получить данные с высоким покрытием в интересующих областях генома, был разработан метод целенаправленного секвенирования (Gabrieli Т. et al., 2018).
В последнее время в области обогащения наблюдается растущий интерес к Cas9-опосредованному нанопоровому секвенированию (NCATS) (Gilpatrick Т. et al., 2020). Суть метода состоит в создании целенаправленных разрезов участков высокомолекулярной геномной ДНК с помощью активного рибонуклеопротеинного комплекса Cas9-gRNA (РНП) (Sternberg S. Н. et al., 2014). Дефосфорилированная ДНК подвергается избирательному лигированию адаптеров к свежесрезанным участкам, созданными РНП-комплексом. Синтез комплекса Cas9-gRNA происходит по средством предварительно разработанных gRNA, фланкирующих интересующую область.
Такой избирательный метод секвенирования имеет важное значение в решение практических задач при исследовании геномов растений, а также экономически эффективно для идентификации вариантов, особенно с учетом длинных последовательностей с повторяющимися участками. Данный подход позволяет получить высокое покрытие интересующего региона, а также дает возможность полная сборки de novo, что является наилучшим подходом для полной характеристики генетического разнообразия. Охват секвенированием интересующей области имеет решающее значение для изучения закономерностей частоты мутаций и метилирования в различных образцах (Giesselmann P. et al., 2019).
Метод NCATS обеспечивает динамические возможности для оценки геномных перестроек, в том числе больших SVS и трудно отображаемые повторяющиеся регионы. Важно отметить, что, поскольку нанопоровое секвенирование исследует цепочку ДНК, а не секвенирование путем амплификации, возможно одновременно выявить метилирование в этих локусах, обеспечивая биологическое, а также диагностическое понимание эпигенома. Данный способ считывание позволяет легко переходить от метилирования к различным вариациям аллелей, что способствует исследованию специфических эпигенетических изменений.
Сущность изобретения
Задачей изобретения, является разработка нового способа одновременного нанопорового секвенирования полных последовательностей значимых генов твердой пшеницы с помощью рибонуклеопротеиновых комплексов Cas9/гPHK.
Для экстракции геномной ДНК твердой пшеницы ЦТАБ-методом (Kirov I. et. al., 2021) используются четырехдневные проростки. Оценка качества выделенной геномной ДНК проводится путем гель-электрофореза в 1,5% агарозном геле при напряжении ЗВ/см. Концентрация и чистота геномной ДНК оценивается на приборах NanoDrop One UV-Vis Spectrophotometer (Thermo Scientific, USA) и Quantus Fluorometer (Promega, USA). Для дальнейших исследований подходит только чистая ДНК (А260/А280 ~ 1,8 и А260/А230 ~ 2,0 по данным NanoDrop) практически без различий в концентрациях, полученных с помощью приборов Nanodrop и Quantus.
Далее проводится обогащение геномной ДНК высокомолекулярными фрагментами с помощью набора Short Read Eliminator XL Kit (Circulomics, США). Образец геномной ДНК доводится до общего объема 60 мкл с концентрацией 150 нг/. Далее к образцу добавляется 60 мкл буфера SRE XL и центрифугируется при 10000 g в течение 30 минут при комнатной температуре. После удаления надосадочной жидкости осадок промывается 200 мкл 70% этилового спирта с дальнейшим центрифугированием при 10000 g в течение двух минут при комнатной температуре. Полученный осадок растворяется 50 мкл буфера ЕВ.
Для проектирования гРНК применяется программа FlashFry с инструментом «discover» и эталонными последовательностями генома. Осуществляется поиск всех позиций гРНК, подобранных на геном. Затем эти гРНК отфильтровываются с помощью функции scoringMetrics с использованием ряда параметров, основным лимитирующим фактором которого является минимизация off-target последовательностей. Далее проводится оценка консервативности позиций гРНК среди геномов разных сортов твердой пшеницы при помощи картирования подобранных гРНК на известные последовательности разных сортов твердой пшеницы. Все гРНК разрабатываются для нацеливания на гомеологичные гены, расположенные как в А, так и в В геномах.
Синтез гРНК осуществляется на ДНК-темплейтах путем ПЦР четырех частично перекрывающихся олигонуклеотидов: специфичного участку целевого гена крРНК, универсальной тpaкpRNA, которая связывается с белковой последовательностью Cas-9, а также двух праймеров Т7 F и Т7 R, являющихся промотором и терминатором для Т7 РНК полимеразы, используемой для in vitro транскрипции. Синтез ДНК-темплейтов для каждой реакции проводится в следующей реакционной смеси: 2 мкл уникального олигонуклеотида крРНК (1 мкМ); 2 мкл тракрРНК (1 мкМ); 2 мкл прямого Т7 и 2 мкл обратного праймеров (по 100 мкМ каждого); 2 мкл 50х dNTP (10 мкМ); 10 мкл 10-кратного буфера Encyclo (Евроген, Москва, Россия); 1 мкл энциклополимеразы (Евроген, Москва, Россия); 79 мкл воды, свободной от РНКаз. Олигонуклеотиды амплифицируются в соответствии с программой ПЦР: начальная денатурация при 98°С (2 мин); 30 циклов денатурации при 98°С (30 сек), отжига при 60°С (30 сек), элонгации при 72°С (30 сек); финальная элонгация при 72°С (1 мин). Затем проводится in vitro транскрипция при 37°С в течение двух часов с использованием набора для высокоэффективного синтеза РНК in vitro (Биолоабмикс, Новосибирск, Россия) согласно протоколу производителя.
Подготовка ДНК библиотеки для Саз9-целевого нанопорового секвенирования на приборе MinlON (Oxford Nanopore Technologies, Великобритания) осуществляется по протоколам, описанным в статье Kirov I. et. al., 2021 (Фиг. 1). Сначала анализируемая ДНК дефосфолирируется ферментом Quick CIP (New England Biolabs, Великобритания), затем дефосфолирированная ДНК расщепляется с помощью рибонуклеопротеидных комплексов Cas9/гPHK. Затем на концы ДНК, свободные от рибонуклеопротеидных комплексов Cas9/гPHK лигируются белковые адаптеры для секвенирования ((Oxford Nanopore Technologies, Великобритания) с помощью набора SQK-LSK109 (Oxford Nanopore Technologies, Великобритания). Секвенирование происходит на приборе MinlON (Oxford Nanopore Technologies, Великобритания), с проточными ячейками R9.4.1 или R10.3.
Далее проводится биоинформатический анализ полученных данных: сборка и выравнивание на геном.
Предложенный метод показывает возможность применения секвенирования nCATS для определения генов фотопериода твердой пшеницы.
Краткое описание чертежей
Фигура 1. Подготовка библиотеки для таргетного нанопорового секвенирования.
Фигура 2. Электрофорез ДНК сортов твердой пшеницы. 1 - Кошелевская; 2 - Киприда; 3 - Кермен; 4 - Золотко; М - маркер размера молекулярной массы Step 100 (Biolabmix, Новосибирск, Россия).
Фигура 3. Измерения концентрации высокомолекулярной ДНК на Qubit 4 и Nanodrop Опес.
Фигура 4. Расположение гРНК относительно исследуемых последовательностей генов яровизации и чувствительности к фотопериоду.
Фигура 5. Последовательности спроектированных гРНК.
Фигура 6. Разделение индивидуальных гРНК на 2 пула
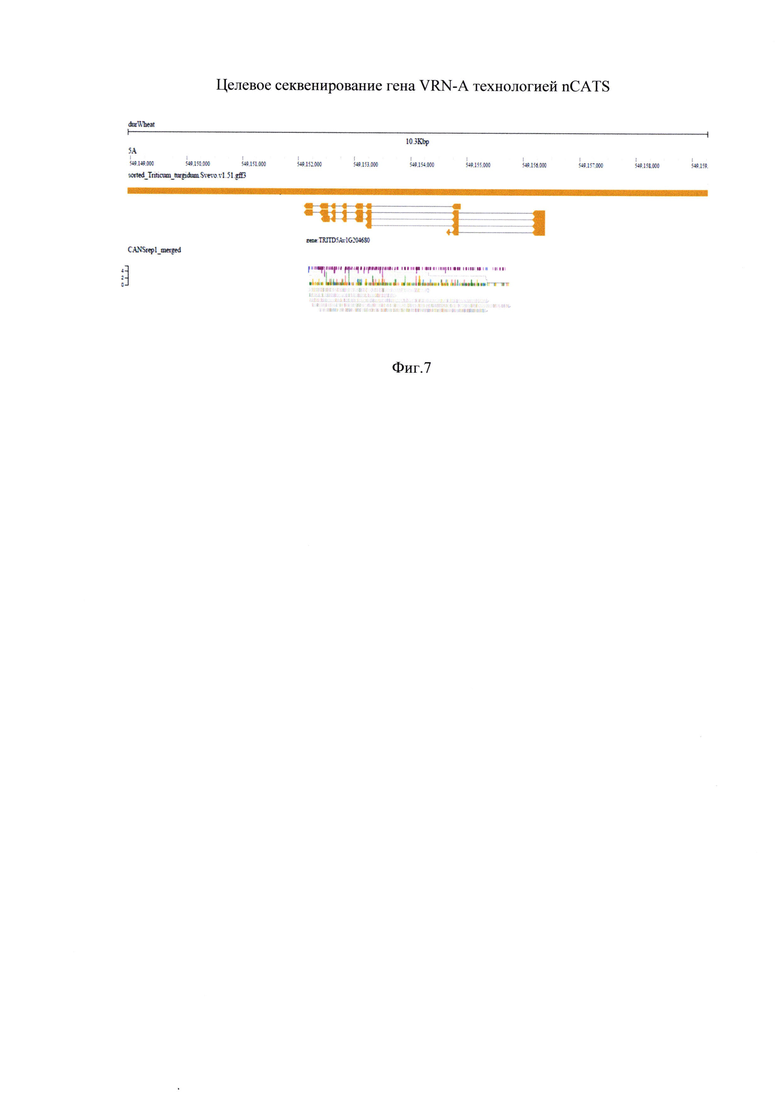
Фигура 7. Целевое секвенирование гена VRN-A технологией nCATS
Фигура 8. Целевое секвенирование гена VRN-B технологией nCATS
Фигура 9. Целевое секвенирование гена Ppd-A технологией nCATS
Пример 1
Определения полных последовательностей значимых генов твердой пшеницы: яровизации (VRN) (требование к яровизации) и фотопериода (Ppd-1) (чувствительность к фотопериоду) методом одновременного нанопорового секвенирования.
У многих растений в ходе эволюции выработались механизмы, определяющие потребность в продолжительном воздействии низкими температурами (яровизации) для последующего перехода к цветению. Это необходимо, чтобы растения переходили к генеративной стадии развития только в подходящее время года. Яровизация генетически контролируется по меньшей мере пятью локусами, VRN-A1, VRN-B1, VRN-D1, VRN4 и VRN-B3. У пшеницы имеется три основных гена яровизации VRN-A1, VRN-B1 и VRN-D1, которые расположены на гомеологичных хромосомах 5А, 5В и 5D соответственно.
У злаковых, в том числе пшеницы, центральным регуляторам фотопериодического ответа является ген Ppd-1, который в свою очередь представлен тремя гомологичными генами, Ppd-A1, Ppd-B1, Ppd-D1, расположенными на коротком плече хромосомы 2.
В качестве объекта были выбран сорт твердой озимой пшеницы: Кошелевская ((Оригинатор/Патентообладатель: селекции ООО «Кошелевский посад», сейчас находится в стадии регистрации и размножения), семена предоставлены Курчатовским геномным центром - ВНИИСБ.
Проращивание семян проводили в чашках Петри на увлажненной фильтровальной бумаге, в 2-х повторностях по 10 семян в каждой чашке Петри. Для выделения высокомолекулярной ДНК отбирались здоровые четырехдневные проростки. Выделение геномной ДНК проводили в вытяжном шкафу ЦТАБ-методом со следующими модификациями. Растительный материал гомогенизировали до порошкообразного состояния при помощи ступок с пестиками с добавлением жидкого азота. К полученному порошку немедленно добавляли предварительно нагретого до 75°С лизирующего буфера (2% СТАВ; 100mM Tris-HCl, рН=8.0; 20mM EDTA, рН=8.0; 1,4М NaCl, 0,25% PVP) в пропорции 500 мкл на 100 мкг растительного материала. Полученную смесь переносили в 1,5 мл пробирки с добавлением 30 мкл р-меркаптоэтанола, далее инкубировали при 75°С в течение 30 минут, периодически переворачивая пробирки. После инкубации к образцам добавляли равный объем смеси (500 мкл) хлороформ:изоамиловый спирт (24:1) для депротеинизации. После центрифугирования при комнатной температуре в течение 30 минут при 13400 g переносили водную фазу в новые 1,5 мл пробирки. Преципитацию комплекса ЦТАБ-ДНК осуществляли добавлением 2 объемов осаждающего буфера (1% СТАВ; 50mM Tris-HCl, рН=8.0; 10mМ EDTA, рН=8.0; 0,125% PVP) с последующим центрифугированием при комнатной температуре в течение 30 минут при 13400 g. После удаления надосадочной жидкости полученный осадок растворяли в 200 мкл 1М NaCl. Преципитацию ДНК проводили с добавлением 200 мкл изопропанола и последующим центрифугированием при комнатной температуре в течение 30 минут при 13400 g. Отмывку полученного осадка и стенок пробирок производили добавлением к осадку 500 мкл 75% этилового спирта и центрифугированием при комнатной температуре в течение 10 минут при 13400 g. После удаления спирта для испарения его остатков пробирки выдерживали открытыми в течение 2 минут. Осадок растворяли в 100 мкл буфера для элюции (10mМ Tris-HCl, рН=8.0; 0,1 mМ EDTA, рН=8.0) с добавлением 1 мкл РНКазы А (10 ед/мкл) к образцу. Полного растворения осадка и элиминации контаминирующей РНК достигали инкубацией в течение 1 часа при 37°С. Далее проводили очистку от РНКазы А добавлением равного объема (100 мкл) смеси хлороформ:изоамиловый спирт и последующего центрифугирования при комнатной температуре в течение 15 минут при 13400 g. Водную фазу переносили в новые 1,5 мл пробирки. Преципитацию ДНК проводили с добавлением 10 мкл 3М ацетат натрия и 100 мкл изопропанола при последующем центрифугировании при комнатной температуре в течение 15 минут при 10000 g. Отмывку осадка и стенок пробирок производили добавлением 500 мкл 80% этилового спирта и дальнейшим центрифугированием в течение 5 минут при 10000 g. Элюцию ДНК проводили в 50 мкл буфера для элюции.
Оценку качества выделенной геномной ДНК проводили путем электрофореза (Фиг. 2). Разделение молекул ДНК производили гель-электрофореза в 1,5% агарозном геле при напряжении 3В/см.
Измерение концентрации и чистоты геномной ДНК проводили при помощи спектрофотометра Nanodrop Опес и флуориметра Qubit 4 (Фиг. 3).
Геномная ДНК проростка твердой пшеницы сорта Кошелевская, ввиду минимального расхождения показателей, полученных при помощи спектрофотометра Nanodrop Опес и флуориметра Qubit 4 идеально подходит для дальнейших исследований.
Обогащение длинными фрагментами осуществляли при помощи Short Read Eliminator Kit (Circulomics, США).
Для аннотации исследуемых генов в геноме твердой пшеницы использовали их известные последовательности, найденные из литературных источников баз данных. Эти последовательности сравнивали с эталонным геномом пшеницы, взятым с базы данных GrainGenes (Triticum turgidum subsp.durum cv. Kronos Earlham Inst, vl scaffolds) с помощью программы BLAST.
Аннотированную последовательность, взятую из геномного браузера использовали для дизайна гРНК (Фиг. 4, Фиг. 5). Гидовые РНК (гРНК) были получены посредством транскрипции in vitro на молекуле двуцепочечной ДНК-матрице, содержащей последовательность Т7-промотора и полученной в результате объединения двух олигонуклеотидов, специфичного и универсального. После синтеза сгРНК осуществлялась сборка рибонуклеопротеинового (РНП) комплекса, представляющего собой комплекс РНК-связывающего белка Cas-9 с рибонуклеиновой кислотой - сгРНК. Рибонуклеопротеиновый комплекс Саs9/крРНК/тракрРНК способен распознавать участки ДНК, идентичные 20 н.п.на 5'-конце крРНК, и нуклеазой Cas9 разрезать обе цепи ДНК с образованием тупых концов.
Для повышения эффективности секвенирования комплекс гРНК был разделен на 2 пула, каждая из которых содержала по одной паре гРНК, фланкирующих последовательность гена (Фиг. 6).
Для транскрипции был использован набор T7-transcription для высокоэффективного синтеза РНК in vitro (Биолабмикс, Новосибирск, Россия) в соответствии с рекомендациями производителя. Анализ концентрации и качества полученных гРНК осуществляли при помощи спектрофотометра Nanodrop OneC (Thermo Scientific, США), Qubit 4 (Thermo Scientific, США) и разделении в 2% агарозном геле. Перед сборкой РНП комплекса проводилась денатурация гРНК для разрушения вторичных структур. Для этого индивидуальные гРНК (50 нг) в каждой из смеси объединяли в молярном соотношении 1:1 в объеме 11 мкл воды свободной от РНКаз, далее инкубировали полученные смеси при 95°С в течение 3 минут и переносили на лед.
Для сборки РНП-комплекса к 11 мкл каждой смеси гРНК добавляли 1 мкл (20 пмоль) EnGen Spy dCas9 (New England Biolabs, Великобритания и 3 мкл (5x) NEB-буфера (New England Biolabs, Великобритания)., полученную смесь инкубировали в течение 30 минут при комнатной температуре.
Подготовка библиотеки для таргетного нанопорового секвенирования состоит из следующих этапов:
1. Дефосфорилирование с помощью фосфатазы 5'-концов фрагментов ДНК в исходном образце. Дефосфорилирование ДНК производилось путем ее обработки щелочной фосфатазой (CIP). Для этого к высокомолекулярной геномной ДНК (концентрация не менее 210 нг/мкл) добавляли 4 мкл буфера CutSmart (10х), 8 мкл Quick CIP и инкубировали 45 минут при 37°С. Инактивацию фермента проводи путем нагревания меси до 80°С в течение 2 минут.
2. Добавление в образец рибонуклеопротеиновых комплексов Cas9/cгPHK/tracrPHK, связывающихся с последовательностями на противоположных концах исследуемых генов.
3. Вырезание целевых генов с 5'-фосфатными группами на концах и аденилирование с помощью Taq ДНК-полимеразы и дАТФ 3'-концов. Белки Cas9 при этом остаются, как правило, связанными с нецелевыми фрагментами, отщепившимися от целевого гена. К дефосфорилированной ДНК добавляли 15 мкл предварительно собранного РНП-комплекса, 1,5 мкл dATP (10 мкМ) и 1 мкл Taq полимеразы. Смесь инкубировали при 37°С в течение 30 мин для внесения разрезов в ДНК, затем 5 мин при 72°С для построения А-хвоста в местах разрезов.
4. Лигирование адаптеров с моторными белками. Поскольку большинство концов нецелевых фрагментов «спрятано» от лигазы в ходе дефосфорилирования и нуклеазами Cas9, то адаптеры в основном лигируются только с целевыми фрагментами. Адаптеры для нанопорового секвенирования (АМХ) были лигированы к расщепленной ДНК Cas9 путем смешивания следующих компонентов: 25 мкл буфера для лигирования LNB (набор LSK109, Oxford Nanopore Technologies, Великобритания), 5 мкл воды сводной от нуклеаз, 12,5 мкл Т4-лигазы ДНК (NEBnext Quick Т4 Ligase) и 5 мкл адаптеров (АМХ). Смесь добавляли к расщепленной ДНК Cas9 и инкубировали в течение 20 мин при комнатной температуре.
5. Очистка на магнитных частицах.
6. Нанесение библиотеки на проточную ячейку и запуск секвенирования. Секвенирование запускали на проточной ячейке MinlON (R9 Version).
Последовательности запусков выполнялись с помощью программного обеспечения MinKNOW (версия 19.2.2).
Заключительная часть процесса секвенирования - биоинформатический анализ полученных последовательностей. В результате определены сиквенсы целевых последовательностей генов. Обогащение аллельного варианта гена яровизации VRN-A составляет 84х (Фиг. 7), аллельная вариация гена яровизации, относящаяся к В геному -VRN В, имеет 118х обогащение (Фиг. 8). Ген чувствительности к фотопериоду Ppd-A имеет наибольшее покрытие из всех изучаемых в исследовании генов, которое составляет 43 8х (Фиг. 9).
Финансирование работ по созданию настоящего изобретения проводилось из средств Соглашения №075-15-2019-1667 от «31» октября 2019 г. о предоставлении из федерального бюджета грантов в форме субсидий в соответствии с пунктом 4 статьи 78.1 Бюджетного кодекса Российской Федерации на осуществление государственной поддержки создания и развития центра геномных исследований мирового уровня «Курчатовский геномный центр» в рамках реализации федерального проекта «Развитие научной и научно-производственной кооперации» национального проекта «Наука».
Список литературы
Lopez-Girona, Е., Davy, М.W., Albert, N.W., Hilario, E., Smart, M.E., Kirk, C,... & Chagne, D. (2020). CRISPR-Cas9 enrichment and long read sequencing for fine mapping in plants. Plant methods, 16(1), 1-13.
De Coster, W., De Rijk, P., De Roeck, A., De Pooter, Т., D'Hert, S., Strazisar, M.,… & Van Broeckhoven, C. (2019). Structural variants identified by Oxford Nanopore PromethlON sequencing of the human genome. Genome research, 29(7), 1178-1187.
Gabrieli, Т., Sharim, H., Fridman, D., Arbib, N., Michaeli, Y., & Ebenstein, Y. (2018). Selective nanopore sequencing of human BRCA1 by Cas9-assisted targeting of chromosome segments (CATCH). Nucleic acids research, 46(14), e87-e87.
Gilpatrick, Т., Lee, I., Graham, J. E., Raimondeau, E., Bowen, R., Heron, A.,… & Timp, W. (2020). Targeted nanopore sequencing with Cas9-guided adapter ligation. Nature biotechnology, 38(4), 433-438.
Sternberg, S.H., Redding, S., Jinek, M, Greene, E.C, & Doudna, J. A. (2014). DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature, 507(7490), 62-67.
Giesselmann, P., Brandl, В., Raimondeau, E., Bowen, R., Rohrandt, C, Tandon, R.,… & Muller, F. J. (2019). Analysis of short tandem repeat expansions and their methylation state with nanopore sequencing. Nature biotechnology, 37(12), 1478-1481.
Ilya Kirov, Pavel Merkulov, Sofya Gvaramiya, Roman Komakhin, Murad Omarov, Maxim Dudnikov, Alina Kocheshkova, Alexander Soloviev, Gennady Karlov, Mikhail Divashuk (2021). Illuminating the transposon insertion landscape in plants using Cas9-targeted Nanopore sequencing and a novel pipeline. bioRxiv - 2021.
Borrelli G.M. et al. Effects of modified processing conditions on oxidative properties of semolina dough and pasta //Cereal Chemistry. - 2003. - T. 80. - №. 2. - C. 225-231. DOI 10.1094/cchem.2003.80.2.225.
Campos К. M. et al. Association of phytoene synthase Psyl-Al and Psyl-Bl allelic variants with semolina yellowness in durum wheat (Triticum turgidum L. var. durum) //Euphytica. - 2016. - T. 207.-№. 1. - C. 109-117. DOI 10.1007/sl0681-015-1541-x.
Cong L. et al. cDNA cloning and expression analysis of wheat (Triticum aestivum L.) phytoene and ζ-carotene desaturase genes //Molecular biology reports. - 2010. - T. 37. - №. 7. - C. 3351-3361. DOI 10.1007/s 11033-009-9922-7.
DONG C. et al. Allelic variation at the TaZds-A1 locus on wheat chromosome 2A and development of a functional marker in common wheat //Journal of Integrative Agriculture. - 2012. - T. 11. - №.7. - C. 1067-1074. DOI 10.1016/S2095-3119(12)60099-9 Muterko A, Kalendar R, Salina E. Novel alleles of the VERNALIZATION 1 genes in wheat are associated with modulation of DNA curvature and flexibility in the promoter region. BMC Plant Biol. 2016;16 Suppl 1:65-81. DOL10.1186/sl2870-015-0691-2.
Muterko A. et al. Discovery, evaluation and distribution of haplotypes and new alleles of the Photoperiod-A1 gene in wheat // Plant molecular biology. - 2015. - T. 88. - №. 1-2. - C. 149-164.
Nakamura S. et al. A wheat homolog of MOTHER OF FT AND TFL1 acts in the regulation of germination // The Plant Cell. - 2011. - T. 23. - №. 9. - C. 3215-3229. DOI: 10.1105/tpc. 111.088492.
Pozniak C. J. et al. Identification of QTL and association of a phytoene synthase gene with endosperm colour in durum wheat // Theoretical and Applied Genetics. - 2007. - Т. 114. - №. 3. - C. 525-537. DOI 10.1007/s00122-006-0453-5.
Zhang X. K. et al. Allelic variation at the vernalization genes Vm-A1, Vm-B1, Vm-Dm, and Vm-B3 in Chinese wheat cultivars and their association with growth habit // Crop science. - 2008. - T. 48. - №. 2. - C. 458-470. DOI:10.2135/cropsci2007.06.0355.
McKenna A, Shendure J. FlashFry: a fast and flexible tool for large-scale CRISPR target design. BMC biology. 2018 Dec;16(l):l-6.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ОПРЕДЕЛЕНИЯ ГЕНОМНОЙ ЛОКАЛИЗАЦИИ И ЧИСЛА КОПИЙ Т-ДНК В ГЕНОМЕ ТРАНСФОРМИРОВАННЫХ РАСТЕНИЙ С ПОМОЩЬЮ CAS9-ЦЕЛЕВОГО НАНОПОРОВОГО СЕКВЕНИРОВАНИЯ | 2021 |
|
RU2785923C1 |
| МОЛЕКУЛА РНК-ПРОВОДНИКА ДЛЯ ГЕНОМНОГО РЕДАКТИРОВАНИЯ ПРОТОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ОДНОДОЛЬНЫХ ЗЕРНОВЫХ С ПРИМЕНЕНИЕМ СИСТЕМЫ CRISPR/CAS9 | 2020 |
|
RU2762831C1 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С НУКЛЕОТИДНОЙ ДЕЛЕЦИЕЙ В ПРОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772577C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С НУКЛЕОТИДНОЙ ВСТАВКОЙ В ПРОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772578C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С БИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В ПРОТОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772575C2 |
| Молекула РНК-проводника sgRNA для внесения мутаций в консервативный участок промоторной области гена PPD-D1 мягкой пшеницы с применением системы редактирования генома CRISPR/Cas9 | 2024 |
|
RU2822358C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА ISA1 У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2833963C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА ISA1 У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2833966C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА GBSSI У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2817377C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА GBSSI У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2834229C1 |








Изобретение относится к области биотехнологии и селекции растений, а именно к способу одновременного секвенирования нуклеотидных последовательностей панели генов, лежащих в основе важных для селекционного процесса признаков твердой пшеницы. Указанный способ включает в себя следующие этапы: выделение высокомолекулярной фракции ДНК из 4-дневных проростков твердой пшеницы (HMW DNA), обогащение геномной ДНК высокомолекулярными фрагментами; проектирование гРНК, специфичных для значимых генов твердой пшеницы, и синтез гРНК in vitro на молекуле двуцепочечной ДНК-матрицы, сборку рибонуклеопротеиновых комплексов Cas9/гРНК, Cas9-целевое нанопоровое секвенирование, биоинформатический анализ полученных данных - сборка и выравнивание на геном. 9 ил., 1 пр.
Способ одновременного нанопорового секвенирования полных последовательностей значимых генов твердой пшеницы, включающий в себя:
выделение высокомолекулярной фракции ДНК из твердой пшеницы (HMW DNA),
обогащение геномной ДНК высокомолекулярными фрагментами;
дизайн гРНК, специфичных для значимых генов твердой пшеницы, и синтез гРНК in vitro на молекуле двуцепочечной ДНК-матрицы,
сборку рибонуклеопротеиновых комплексов Cas9/гРНК,
Cas9-целевое нанопоровое секвенирование,
биоинформатический анализ полученных данных - сборка и выравнивание на геном.
| LOPEZ-GIRONA Е | |||
| et al., CRISPR-Cas9 enrichment and long read sequencing for fine mapping in plants | |||
| Plant methods | |||
| Способ восстановления спиралей из вольфрамовой проволоки для электрических ламп накаливания, наполненных газом | 1924 |
|
SU2020A1 |
| Vol | |||
| Устройство для электрической сигнализации | 1918 |
|
SU16A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Pp | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| GILPATRICK T | |||
| et al., Targeted nanopore sequencing with Cas9-guided adapter ligation | |||
| Nat Biotechnol | |||
| Способ восстановления спиралей из вольфрамовой проволоки для электрических ламп накаливания, наполненных газом | 1924 |
|
SU2020A1 |
| Vol | |||
| Способ сужения чугунных изделий | 1922 |
|
SU38A1 |
| No | |||
| Очаг для массовой варки пищи, выпечки хлеба и кипячения воды | 1921 |
|
SU4A1 |
| Pp | |||
| Подвижная хлебопекарная печь | 1925 |
|
SU433A1 |
| СИСТЕМЫ CRISPR-CAS И СПОСОБЫ ИЗМЕНЕНИЯ ЭКСПРЕССИИ ПРОДУКТОВ ГЕНОВ | 2013 |
|
RU2687451C1 |
| MALMBERG M.M | |||
| et al., Assessment of | |||
Авторы
Даты
2022-12-15—Публикация
2021-12-07—Подача