Изобретение относится к области медицины, а именно к инфекционным болезням, и может быть использовано для определения риска развития тяжелого течения COVID-19 на основании данных генетического теста. Изобретение позволяет выявлять лиц с повышенным риском развития тяжелого течения новой коронавирусной инфекции 2019 при проведении превентивного генетического тестирования с целью раннего начала профилактических и лечебных мероприятий.
Коронавирусная болезнь 2019 (COVID-19) - инфекционное заболевание, вызываемое SARS-CoV-2, которое стремительно распространилось по земному шару, начиная с февраля 2020 года. SARS-CoV-2 представляет собой вирус одноцепочечной РНК с положительным смыслом в семействе Coronaviridae. Это седьмой известный коронавирус, способный заражать людей, после HCoV-229E, HCoV-OC43, HCoV NL63, HKU1, MERS-CoV и оригинального SARS-CoV. Первые четыре обычно вызывают нелетальные легкие заболевания верхних дыхательных путей, тогда как последние два, а также SARS-CoV-2 могут вызывать тяжелые летальные респираторные заболевания [1], [2], [3].
С начала эпидемии COVID-19 в Ухане исследователи в различных странах пытались определить специфические характеристики вируса SARS-CoV-2 и организма-хозяина, которые бы объясняли разнообразную клиническую картину этой инфекции. Клинические проявления COVID-19 варьируются от бессимптомной инфекции, легкого заболевания верхних дыхательных путей до вирусной пневмонии, которая в тяжелых случаях может прогрессировать до дыхательной недостаточности и смерти [4]. В Российской Федерации активно предпринимаются меры по скринингу, профилактике и клинической стратификации групп риска среди населения с целью снижения распространения инфекции и своевременного лечебно-профилактического вмешательства. С момента начала пандемии по настоящее время в России было зарегистрировано почти 13 млн случаев заболевания и 336 тыс смертей в результате Covid-19 [5]. Для предотвращения смертельных случаев необходимо максимально рано начинать профилактику данного заболевания в группах повышенного риска.
Многочисленные полногеномные ассоциативные исследования (GWAS) продемонстрировали связь между генетической архитектурой хозяина и уязвимостью к последствиям COVID-19. Согласно данным, полученным на российской популяции, локус, достигший полногеномной значимости, расположен в хромосоме 3 рядом с кластером генов LZTFL1, FYCO1, XCR1 и хемокинов в регионе 3р21.31, которые играют роль в иммунной функции, вирусной репликации, функционировании бронхиального эпителия. Эти данные подтверждаются несколькими независимыми полногеномными исследованиями на других когортах [6], [7], [8].
Однако отдельно взятых нуклеотидных полиморфизмов недостаточно, для того чтобы оценить суммарный вклад генетических вариантов в формирование фенотипа с высоким риском тяжелого течения COVID-19.
Наиболее близким аналогом можно считать исследование, которое выполнено согласно публикации международной заявки WO2021237292, заявитель "Genetic Technologies Limited", Австралия, штат Виктория, авторы Dite Gillian Sue, Murphy Nicholas Mark, All-Man Richard. Согласно описанию заявки, разработана полигенная шкала рисков (ПШР) для предикции риска развития тяжелого течения COVID-19. Пациенты, клинические данные и биоматериал в данном исследовании были предоставлены UK Biobank, возраст участников от 50 лет. Исследования основывались только на данных собранных UK Biobank, собственные исследования не проводились. Тяжесть течения определялась фактом госпитализации в стационар. Дополнительно к возрасту, учитывались такие клинические факторы риска как индекс массы тела, пол, статус курения, наличие аутоиммунных заболеваний, опухолевых заболеваний, группа крови по системе АВО, наличие диабета и заболеваний респираторной системы (за исключением астмы), артериальная гипертензия.
В нашем исследовании приняли участие 5629 пациентов: 2279 мужчин и 3350 женщин в возрасте от 18 до 95 лет (медианный возраст - 51 год), из числа стационарных и амбулаторных пациентов различных медицинских учреждений Российской Федерации. Участники были разделены на две группы в зависимости от наблюдаемой симптоматики: бессимптомные участники/участники с легким течением заболевания и участники со средней/тяжелой/крайне тяжелой степенью заболевания. В основе деления было отсутствие или наличие у участников вирусной пневмонии.
Диагностику и оценку тяжести заболевания проводили в соответствии с рекомендациями Минздрава РФ. По данным КТ-диагностики у пациентов со средней, тяжелой и крайне тяжелой симптоматикой была диагностирована вирусная пневмония.
В отличие от работы выполненной "Genetic Technologies Limited", критериями тяжести течения заболевания являются объективные данные по КТ, показатели сатурации и выраженность дыхательной недостаточности, температура, стабильность гемодинамики, уровень С-реактивного белка, развитие полиорганной недостаточности, септического шока.
Таким образом, в нашей работе разработана полигенная шкала риска тяжелого течения COVID-19 у российской популяции, с учетом особенностей разнообразия этнических групп и среды обитания.
ПШР могут быть использованы для скрининга и выявления группы риска, что поможет улучшить информирование пациентов. Богатая фенотипическая информация в крупномасштабных генотипированных когортах дает возможность систематически оценивать прогностическую эффективность ПШР по широкому спектру признаков. Подобный подход уже используется для выявления среди населения пациентов с высоким генетическим риском таких полигенных заболеваний как ишемическая болезнь сердца, сахарный диабет, дислипидемия [9], [10] Например, авторы Luanluan Sun et al, используя данные UK Biobank о 306 654 человек без сердечно-сосудистых заболеваний (ССЗ) в анамнезе и не получавших гиполипидемическую терапию, рассчитали полигенную шкалу риска ССЗ. Согласно результатам этой работы, добавление информации о генетическом риске, полученной на основании ПШР, увеличило меру различения риска (С-индекс) на 0,012 (95% ДИ 0,009-0,015) и привело к постоянным чистым улучшениям реклассификации примерно на 10% и 12% в случаях и без случаев, соответственно.
Опираясь на выше представленные положения о целесообразности и актуальности разработки инструмента, который позволит выполнить качественную стратификацию по группам риска клинического течения новой короновирусной инфекции 2019, мы построили полигенную шкалу риска тяжелого течения у пациентов с COVID-19. Обнаруженные нами варианты генов ZNF568, GPR173, PCDH15, IGSF3 вошли в полигенную шкалу риска тяжелого течения COVID-19. Среди опубликованных работ полигенная шкала риска тяжелого течения Covid-19 по результатам GWAS была рассчитана для различных субпопуляций в Индии по данным, полученным в исследовании Prakrithi Р. [11], а также австралийской командой "Genetic Technologies Limited", которая использовала базу данных пациентов UK Biobank. Учитывая генетическую неоднородность различных популяций, наиболее актуальна разработка собственных полигенных шкал риска.
Перечень рисунков:
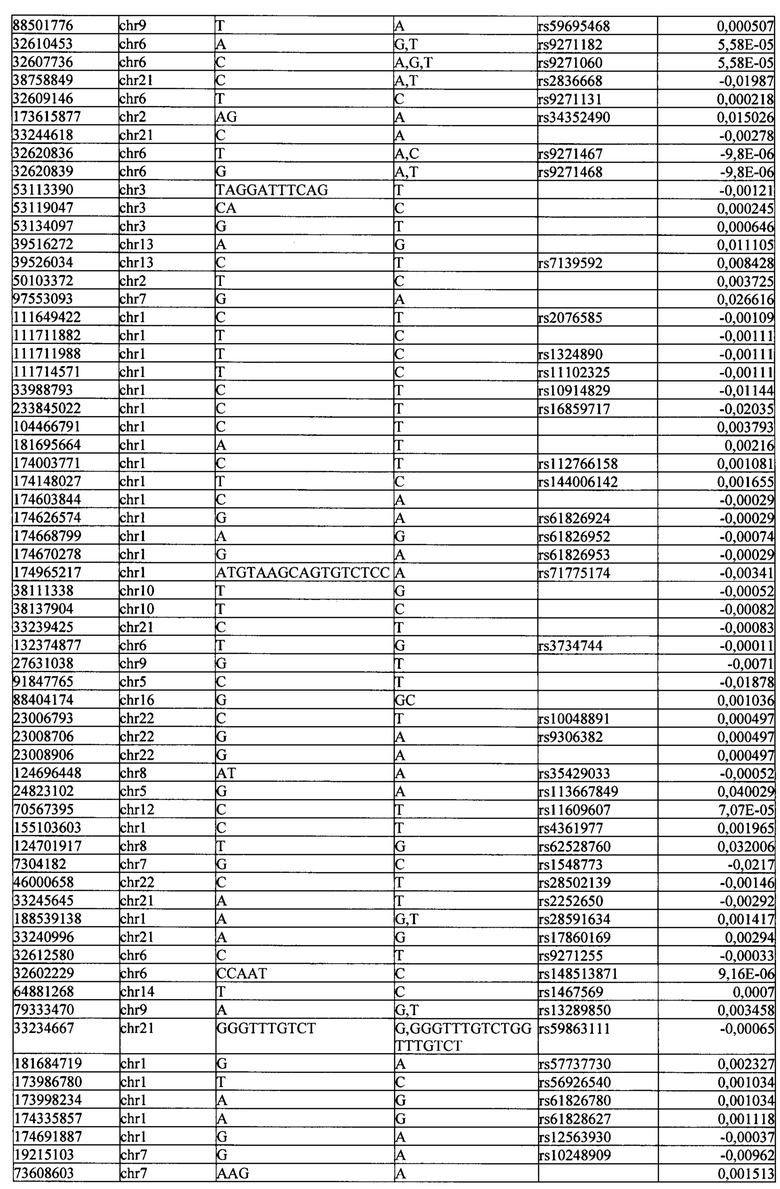
Рисунок 1. Изменения значений точности модели в зависимости от количества анализируемых признаков. Голубым выделен диапазон стандартного отклонения, полученный в результате 10-кратной перекрестной проверки. На рисунке А показана точность при самом высоком значении параметра регуляризации - λ=10, на рисунке В - при λ=1, на рисунке С - при λ=0,1.
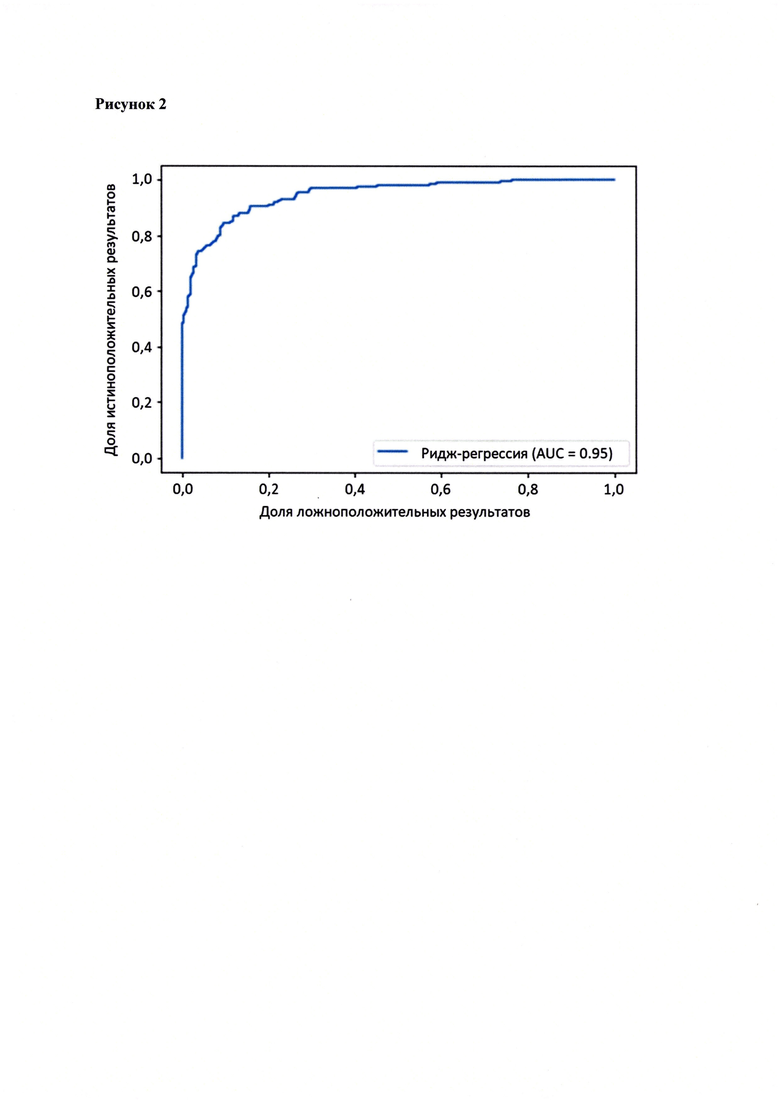
Рисунок 2. ROC-кривая конечной модели на основе ПШР.
Задачей изобретения является расширение возможностей генетической диагностики для оценки риска тяжелого течения COVID-19, а именно создание достоверного способа прогнозирования риска развития тяжелого течения COVID-19 на основании данных генетической диагностики наличия: полиморфизмов генов ZNF568, GPR173, PCDH15, IGSF3 связанных с тяжелым течением SARS-CoV-2 инфекции, и других.
Технический результат изобретения - разработка способа прогнозирования риска развития тяжелого течения COVID-19 на основании генетической диагностики категорий риска тяжелого течения COVID-19, что будет способствовать повышению эффективности прогнозирования клинического течения инфекции и проведению своевременных профилактических мероприятий населению.
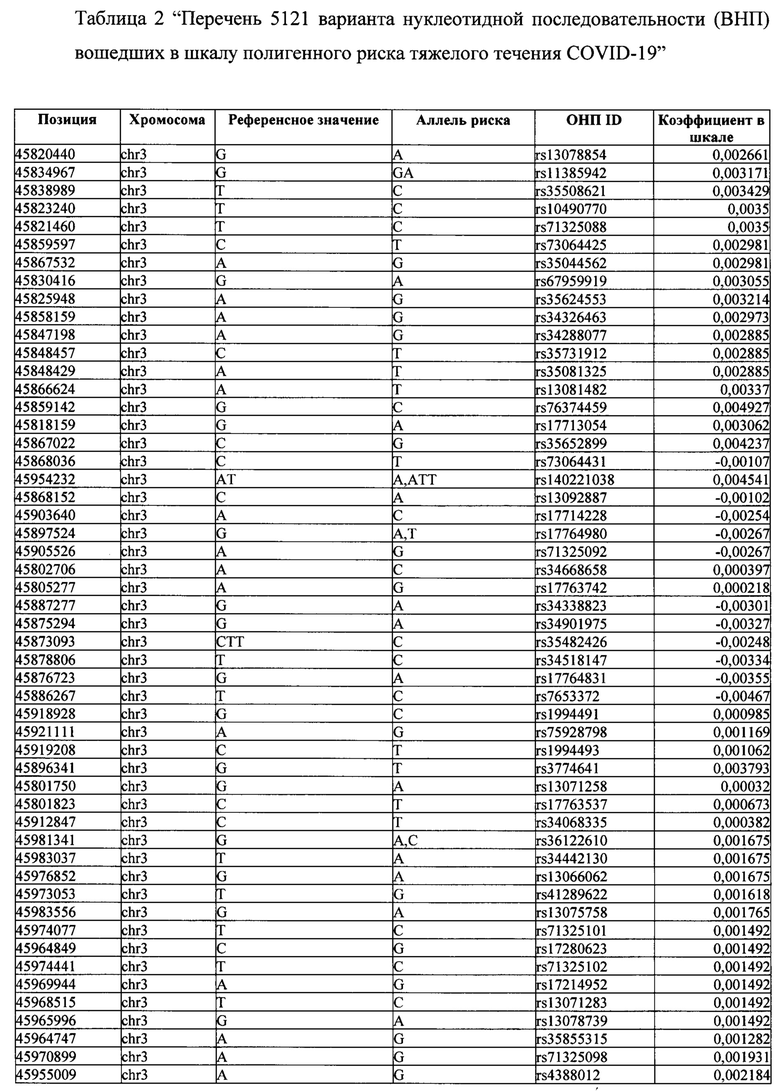
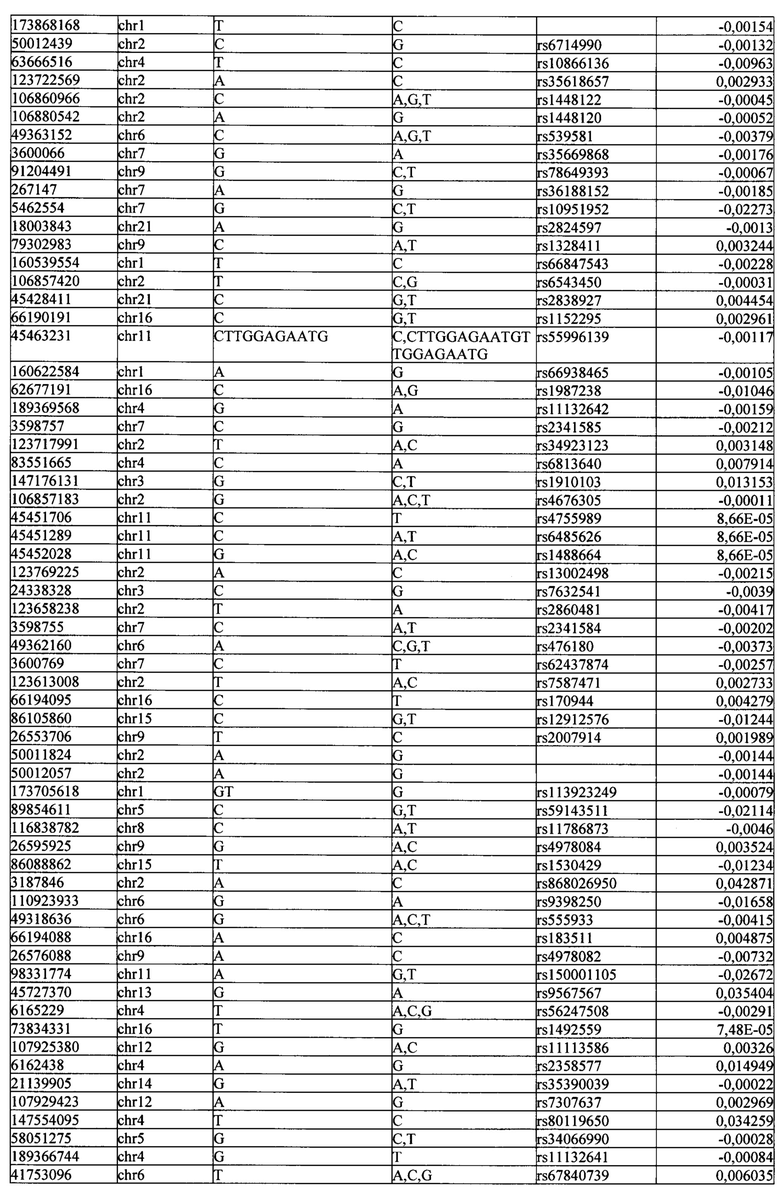
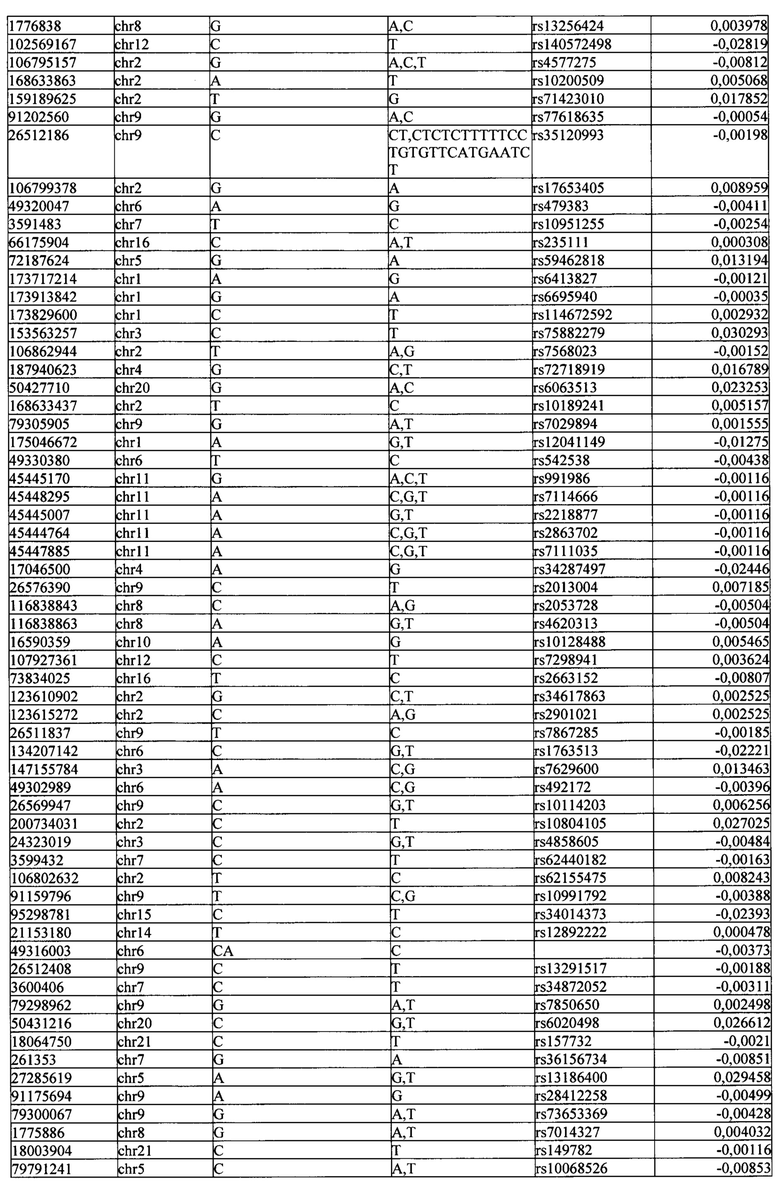
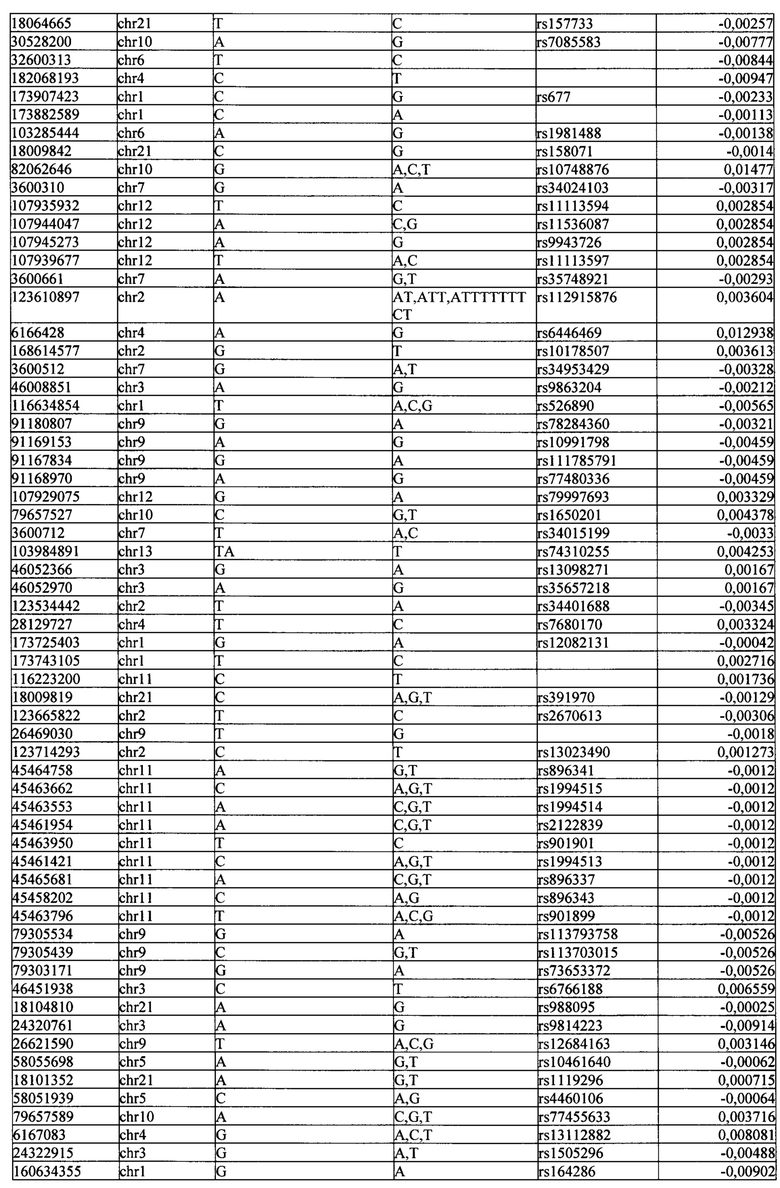
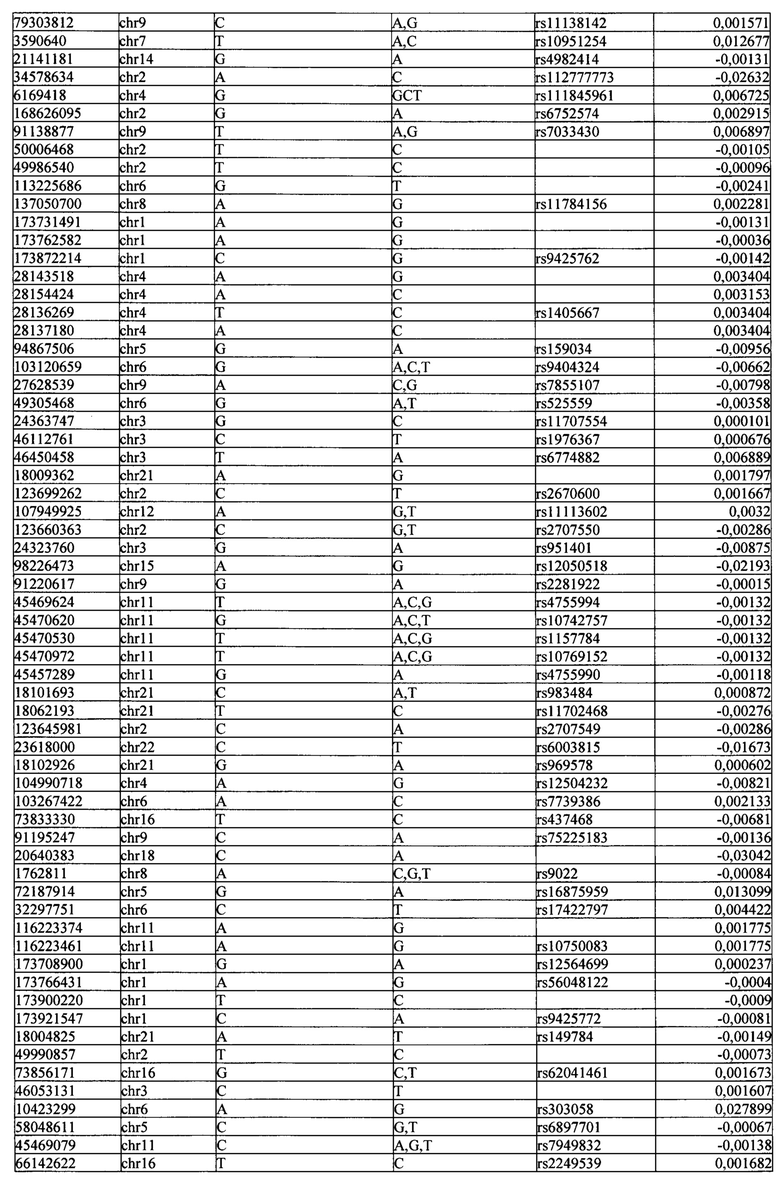
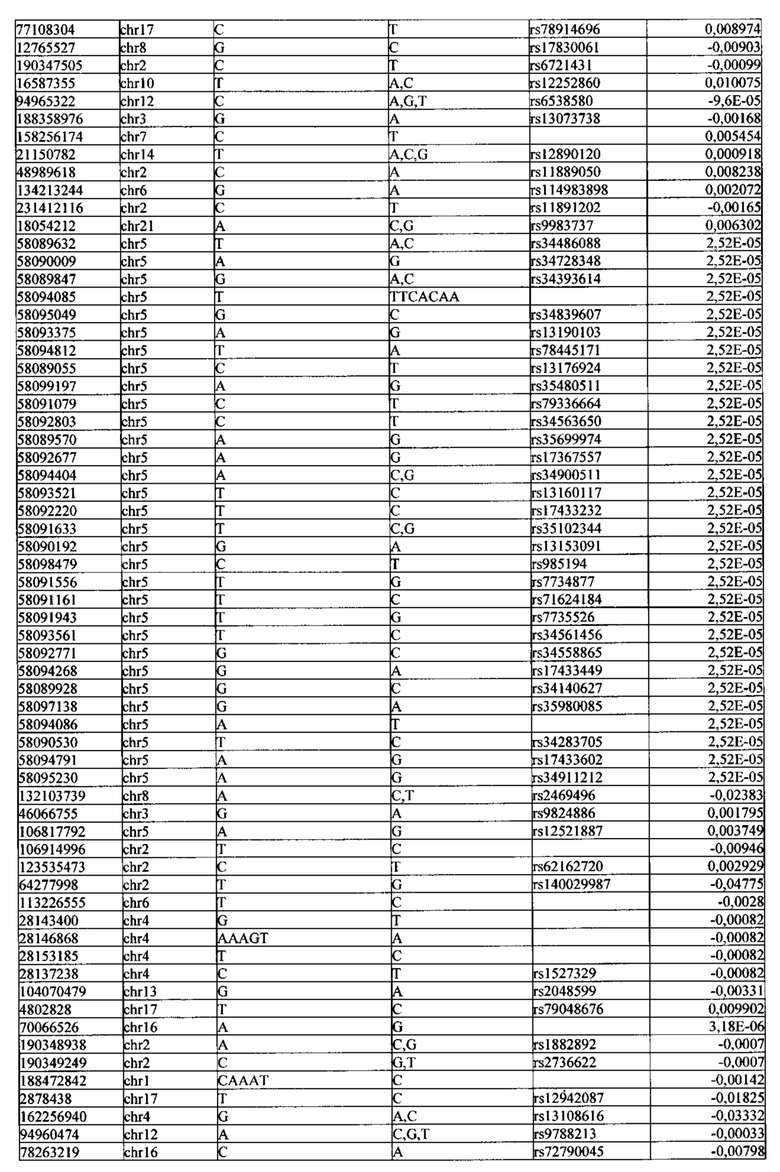
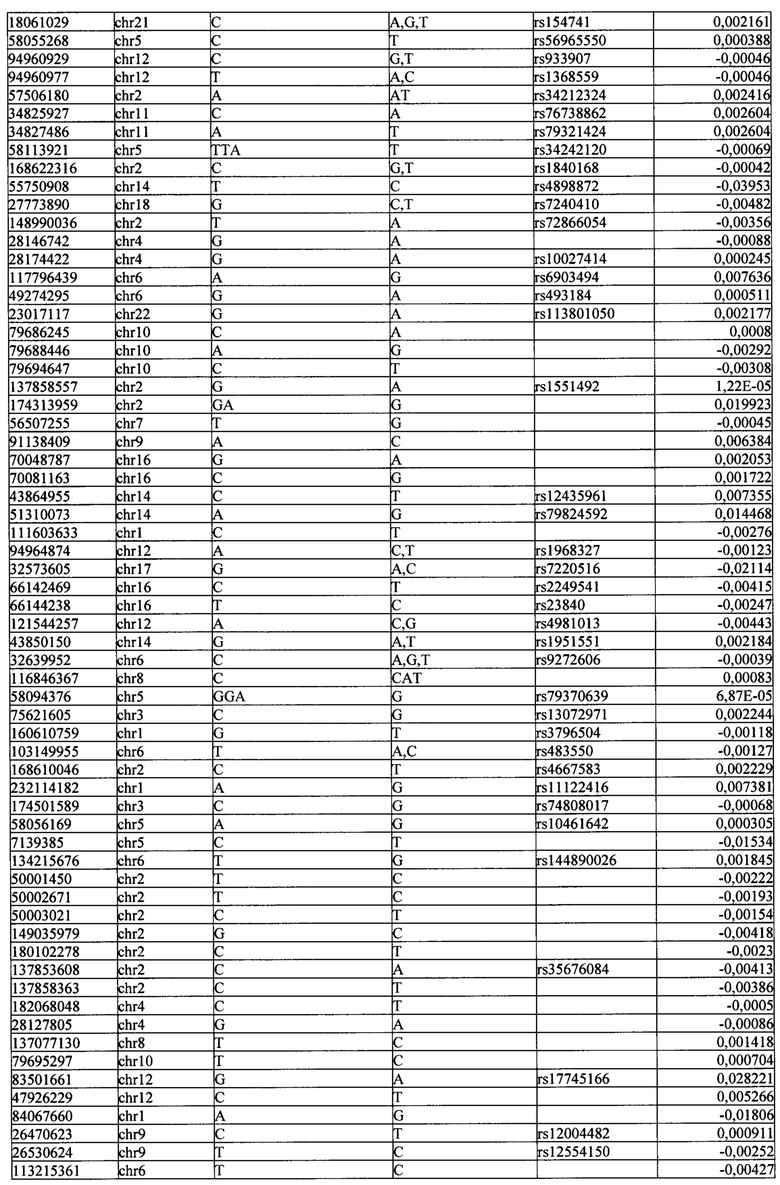
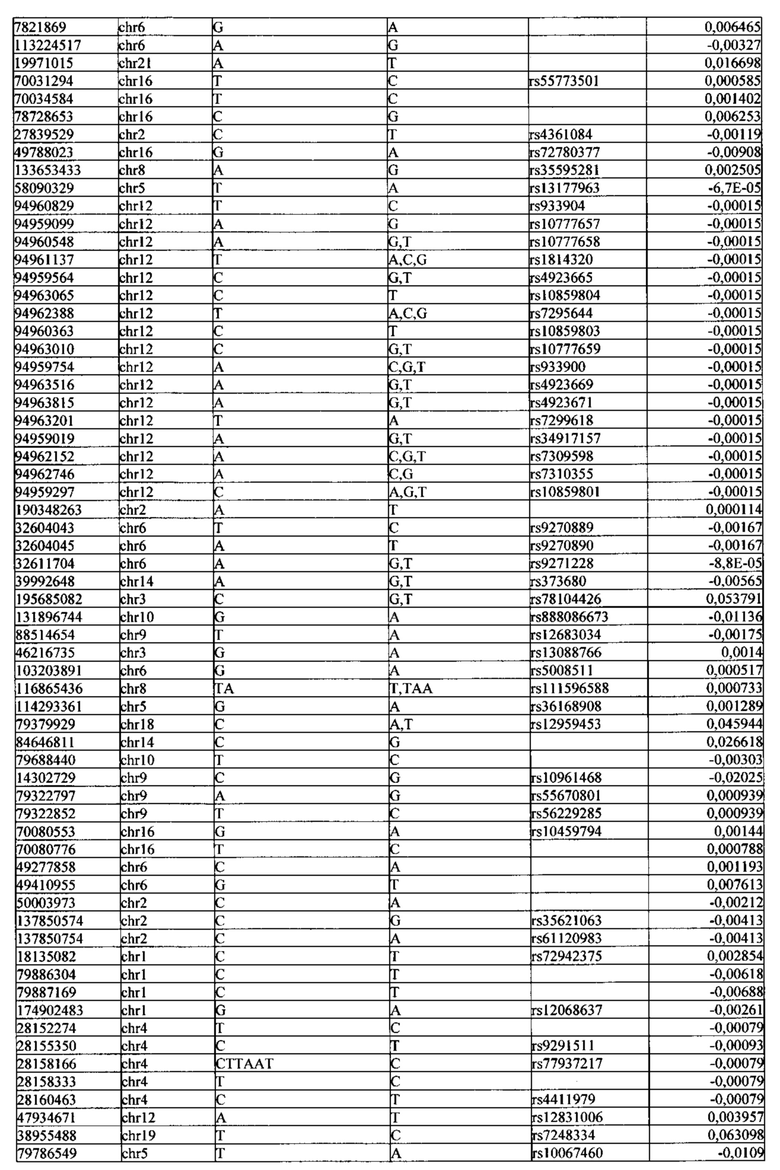
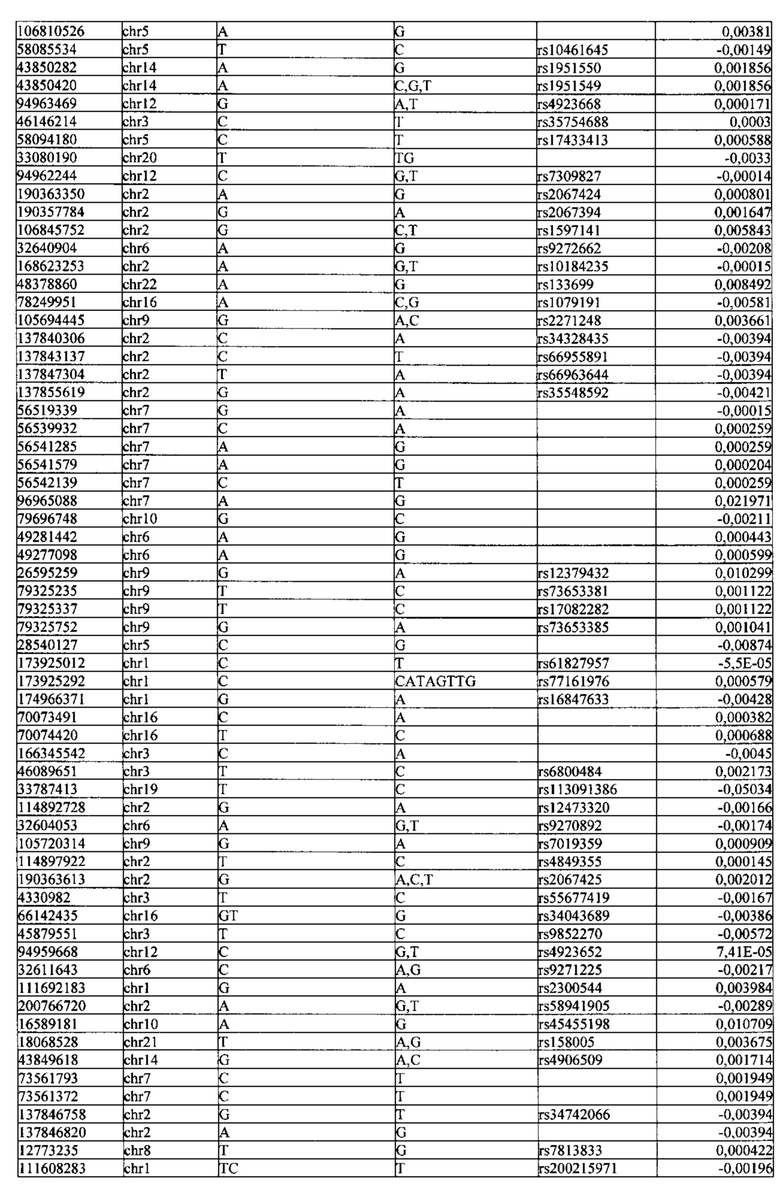
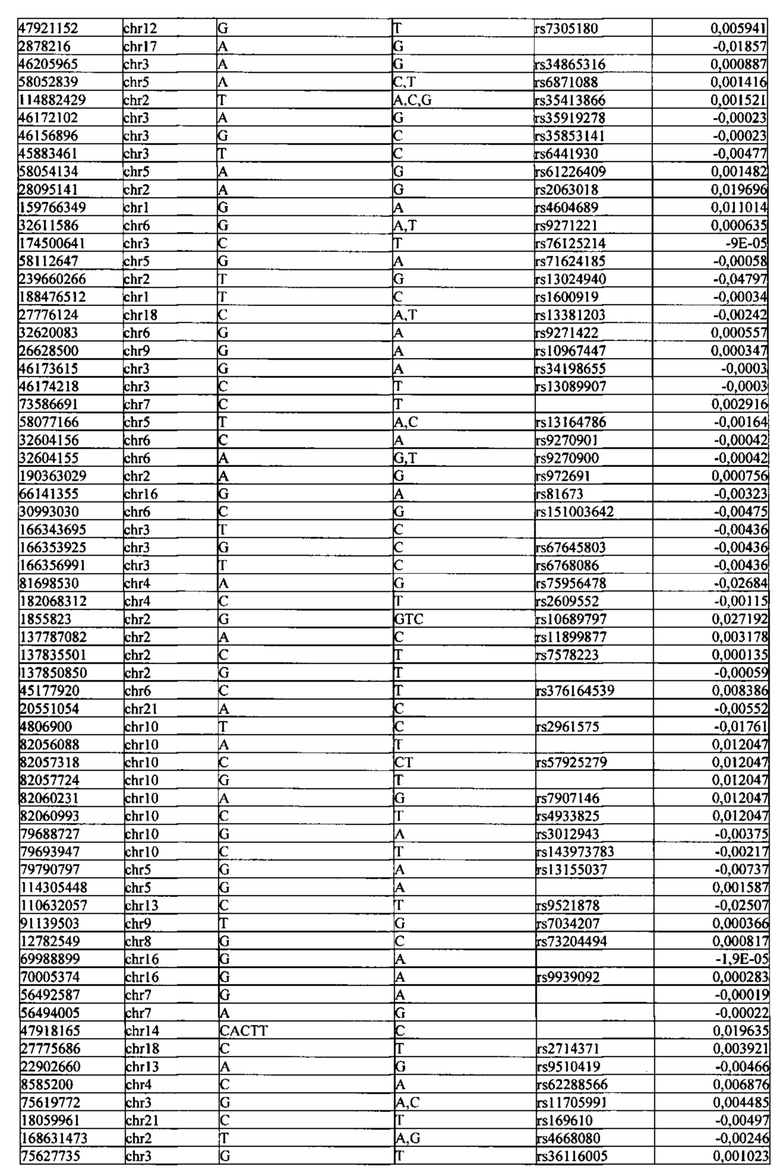
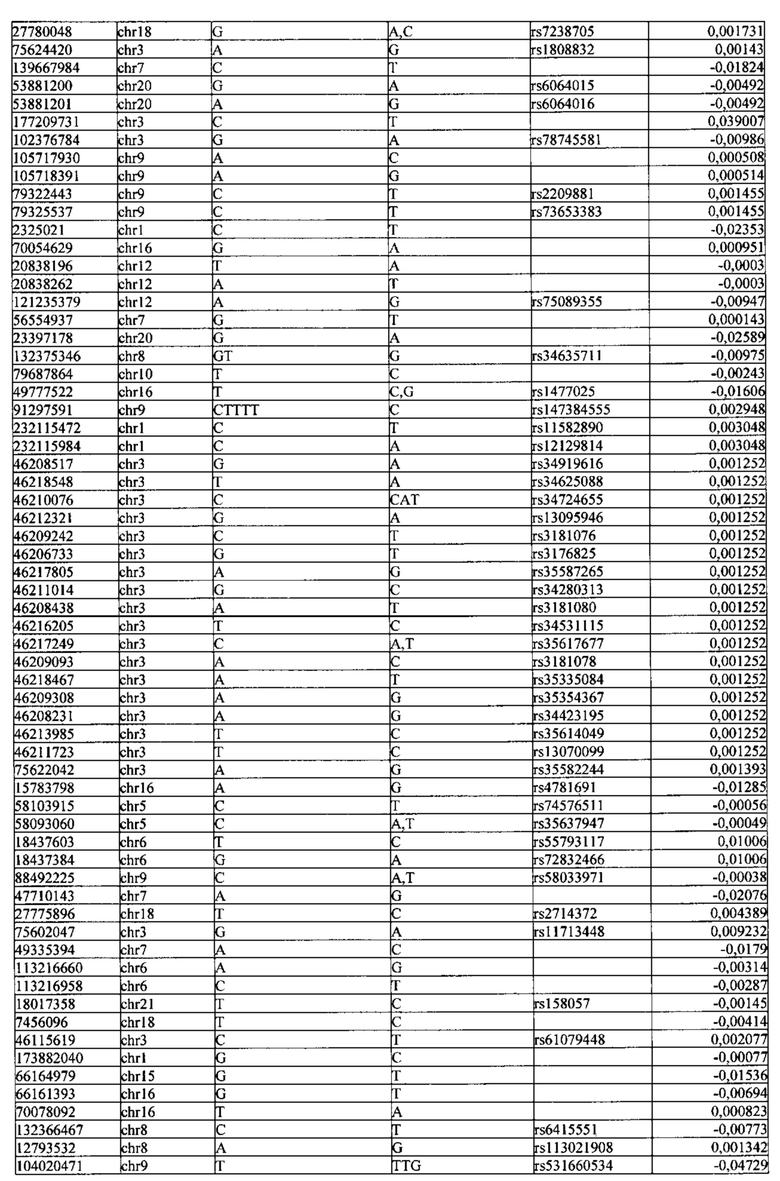
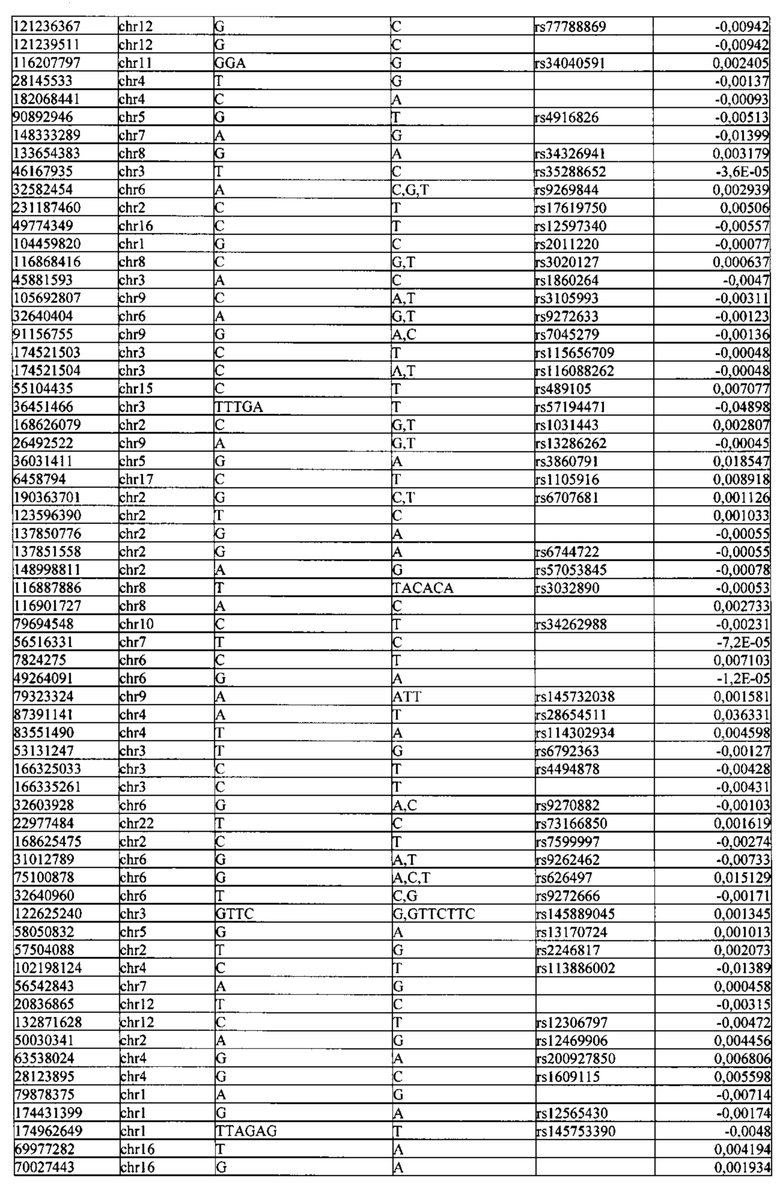
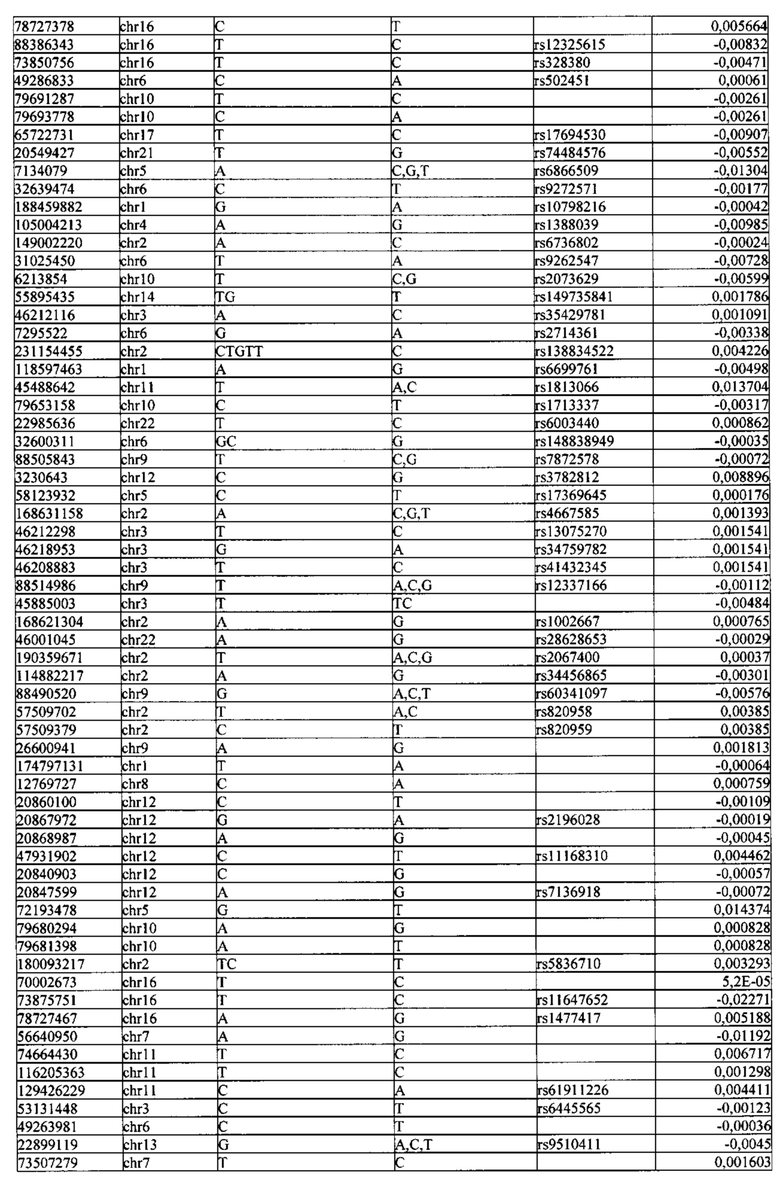
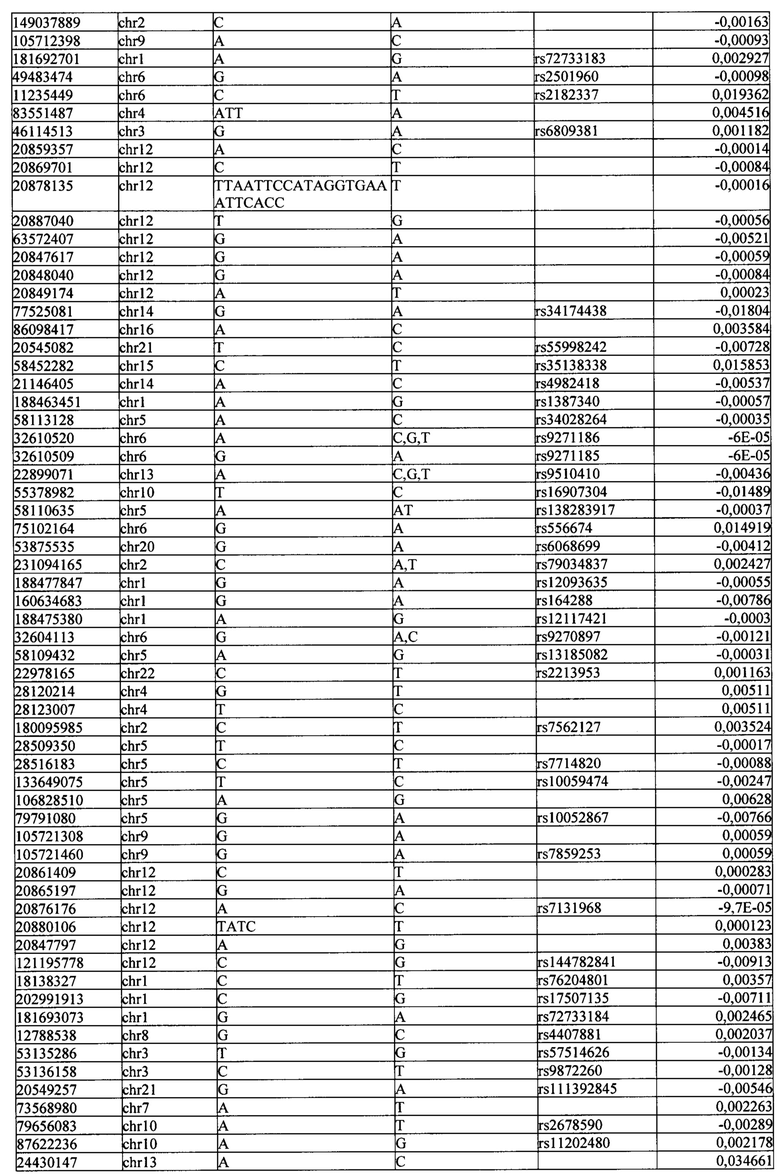
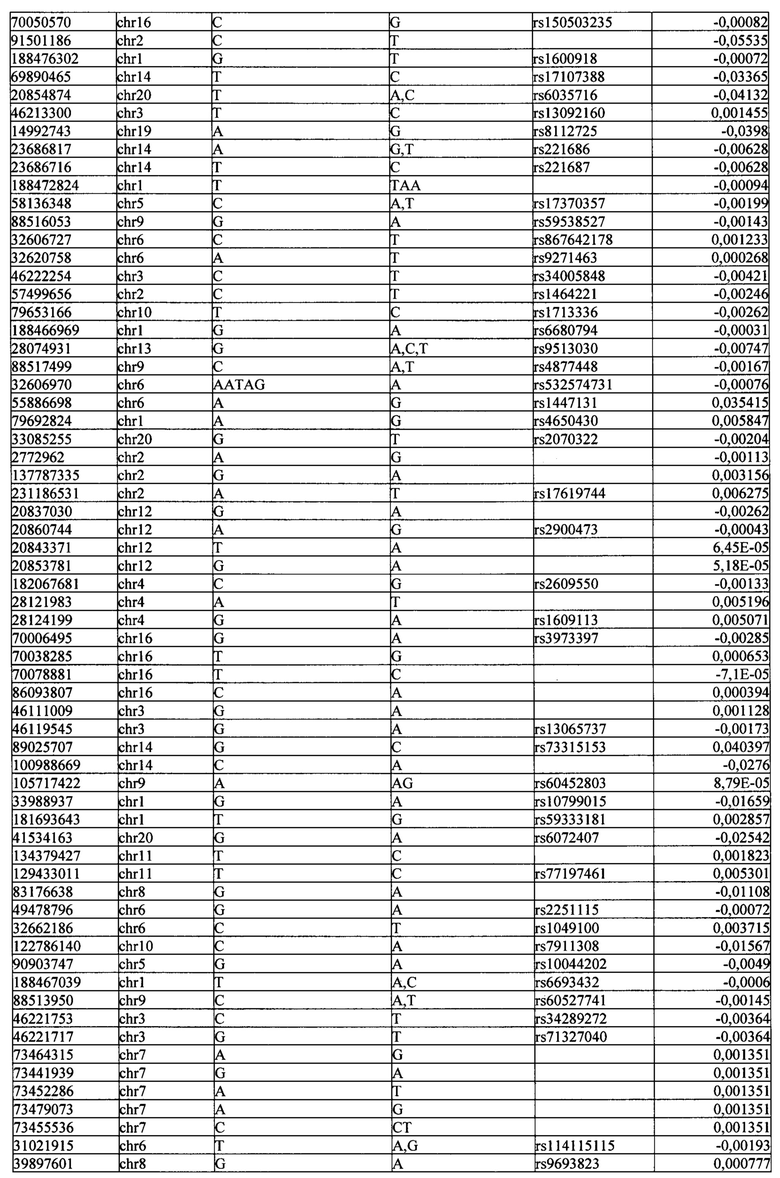
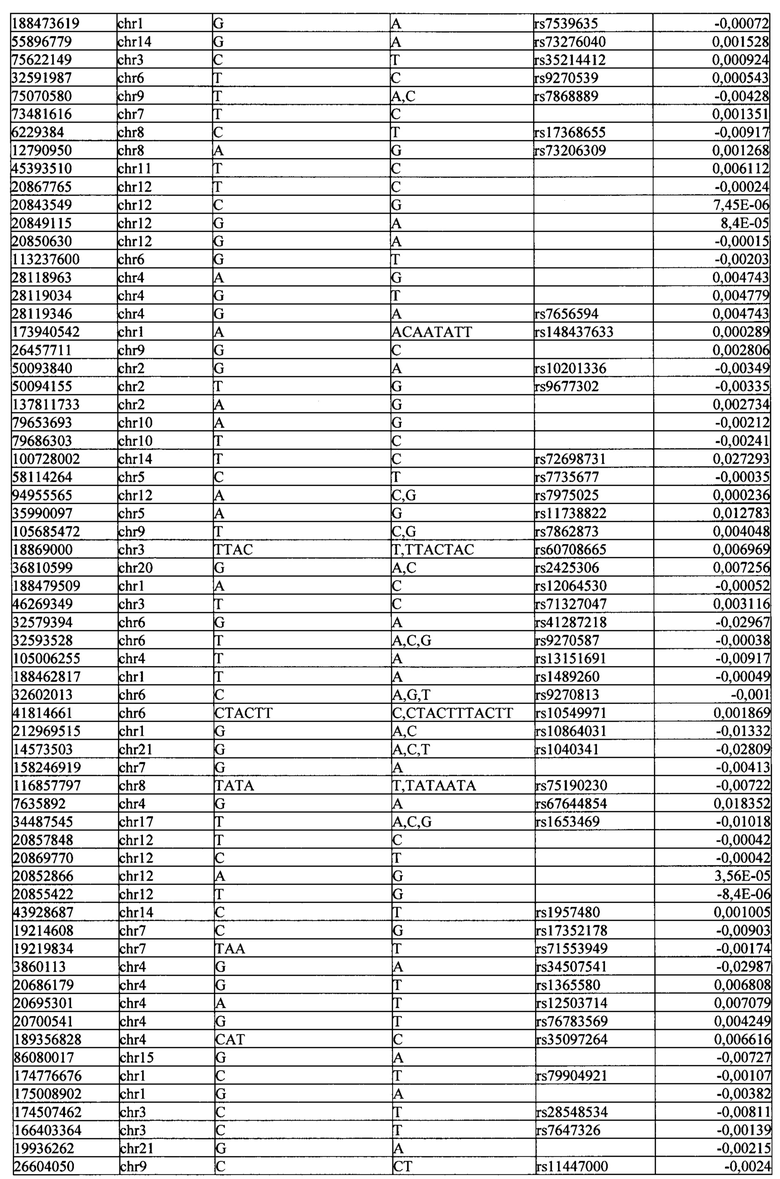
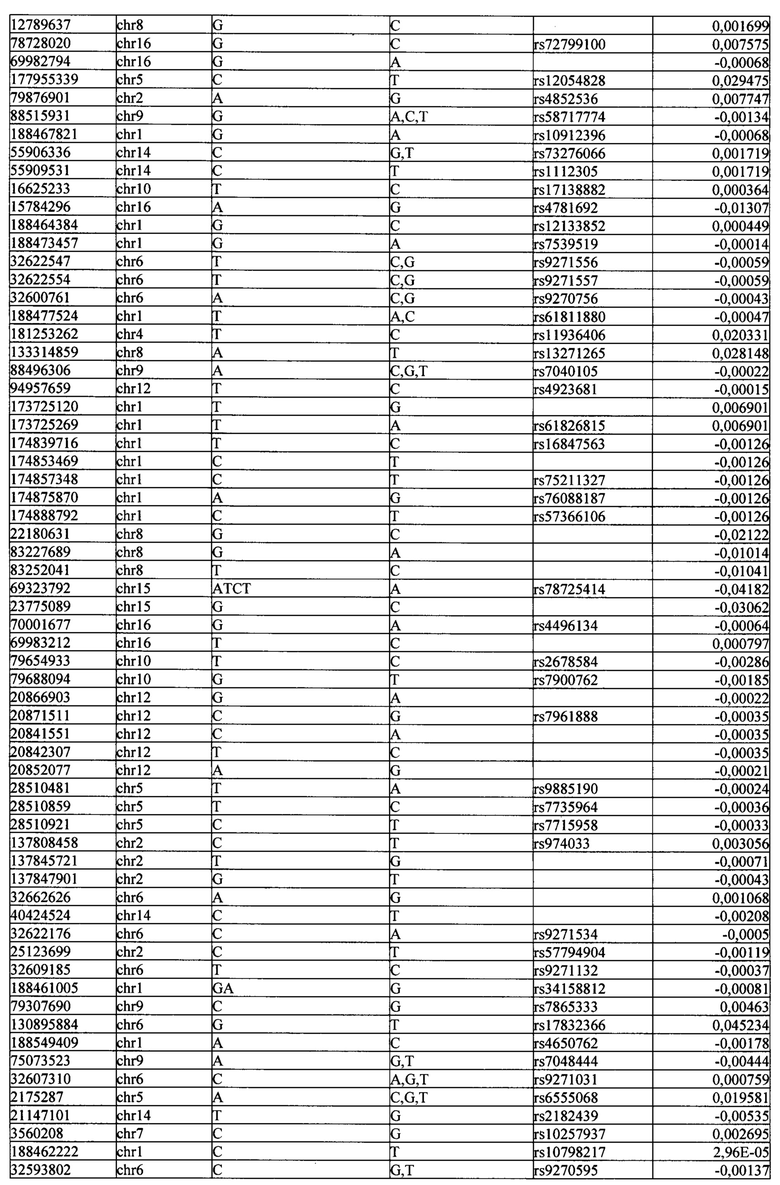
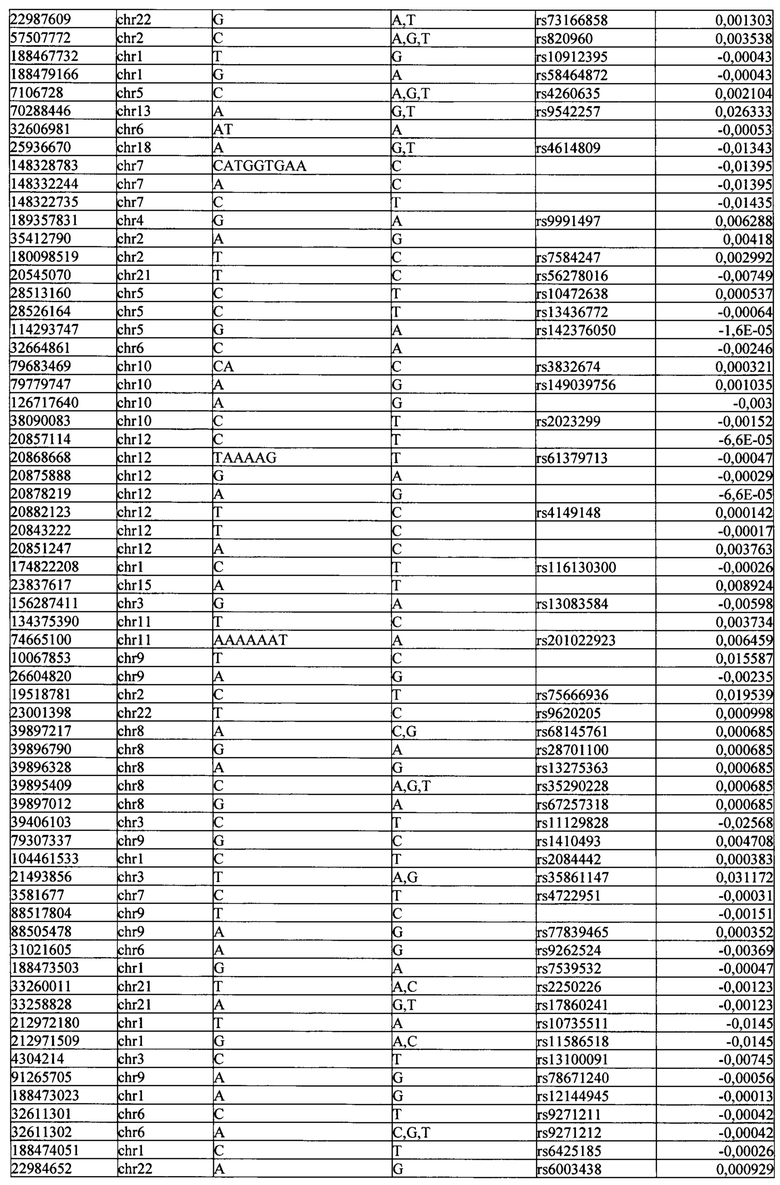
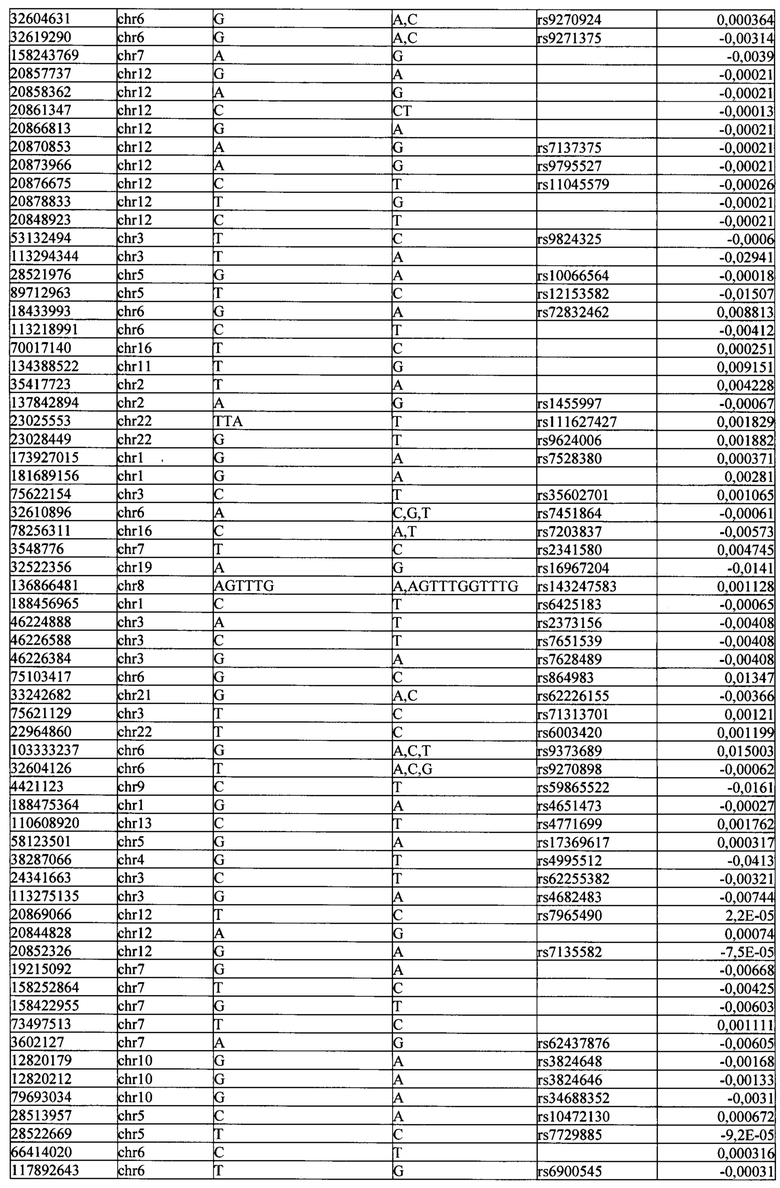
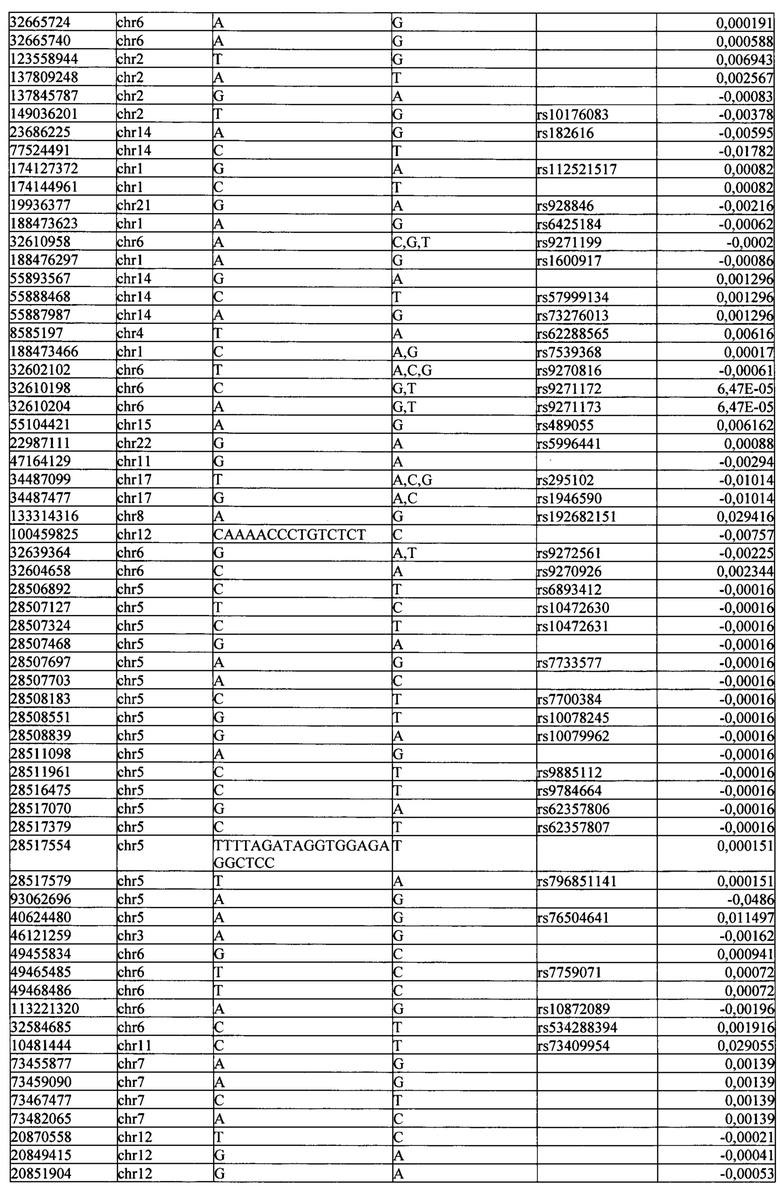
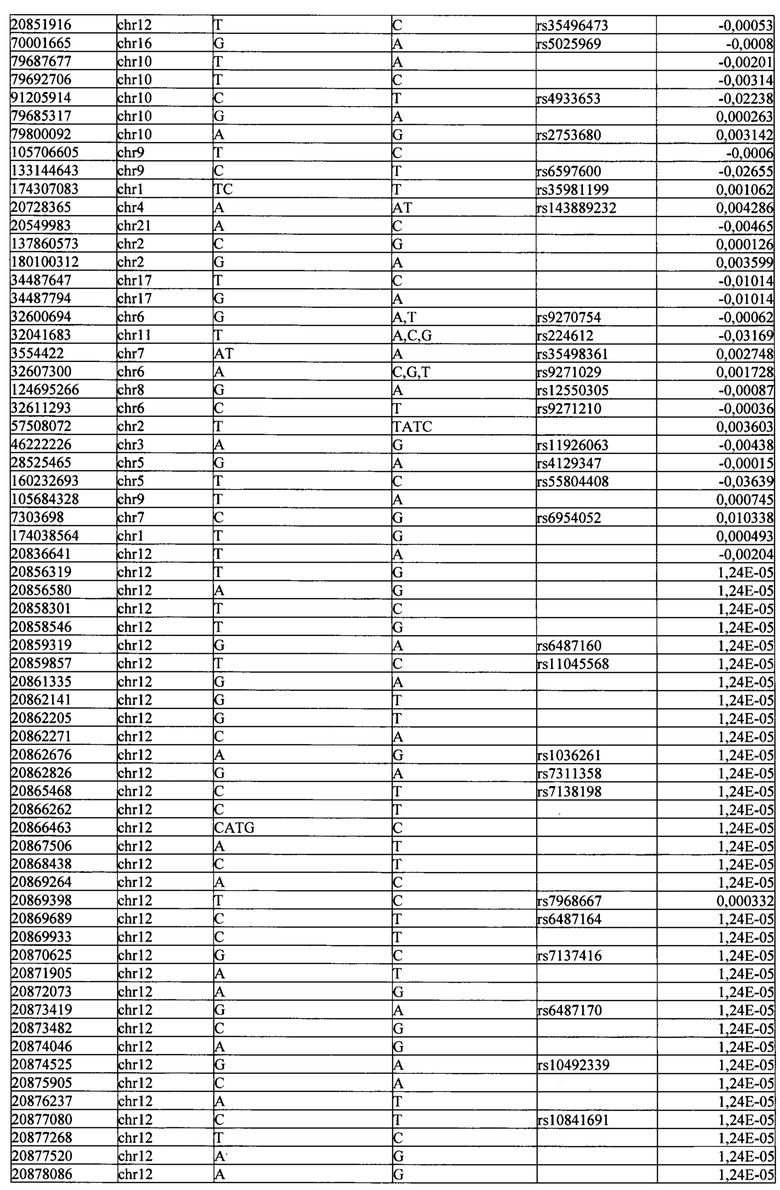
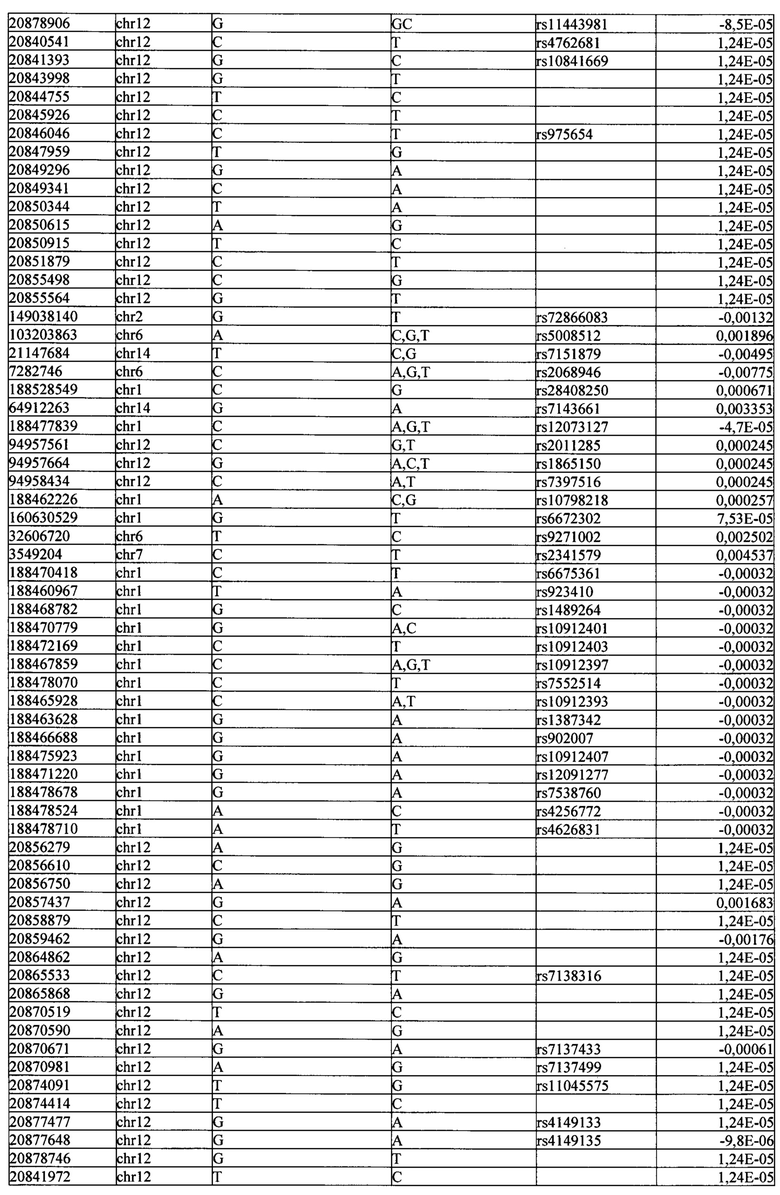
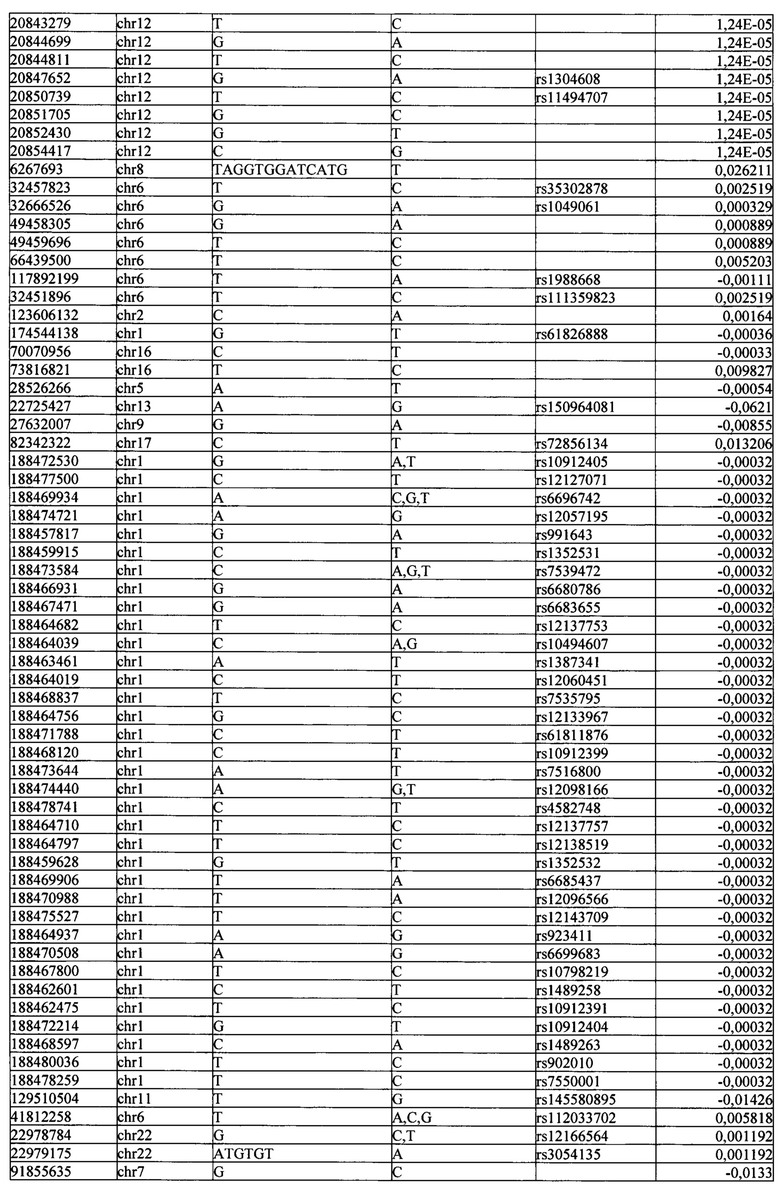
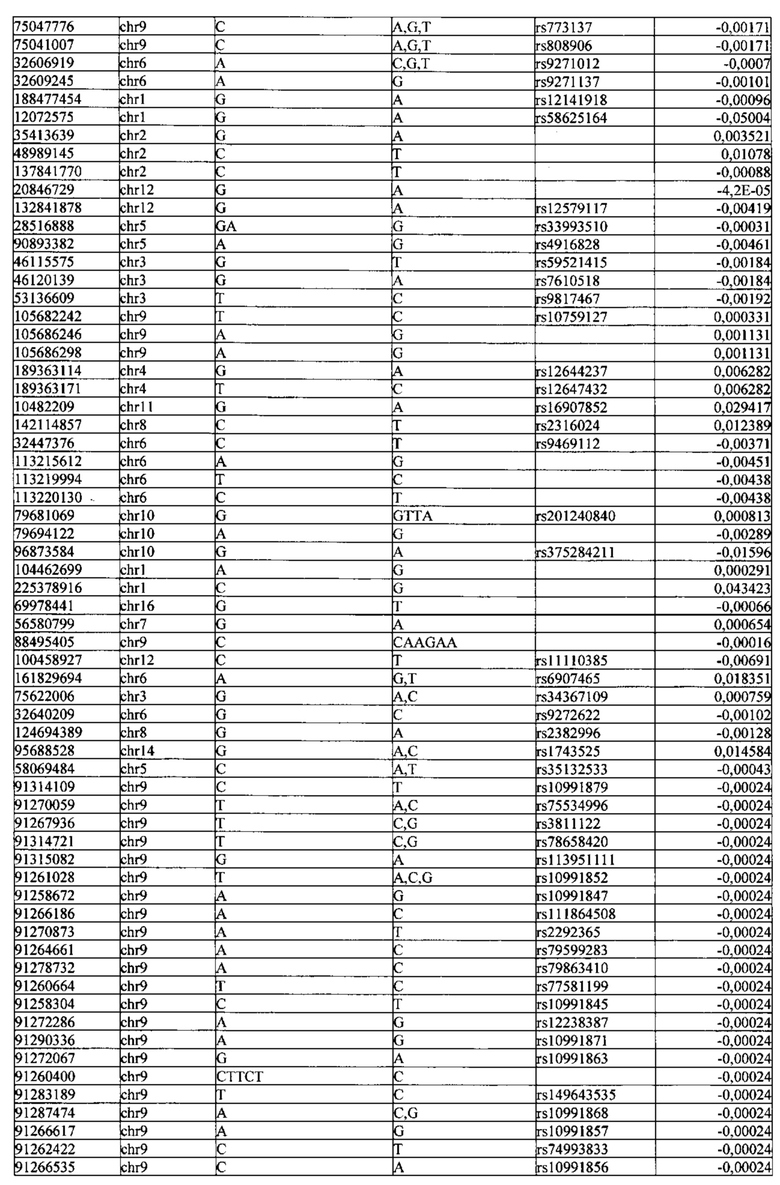
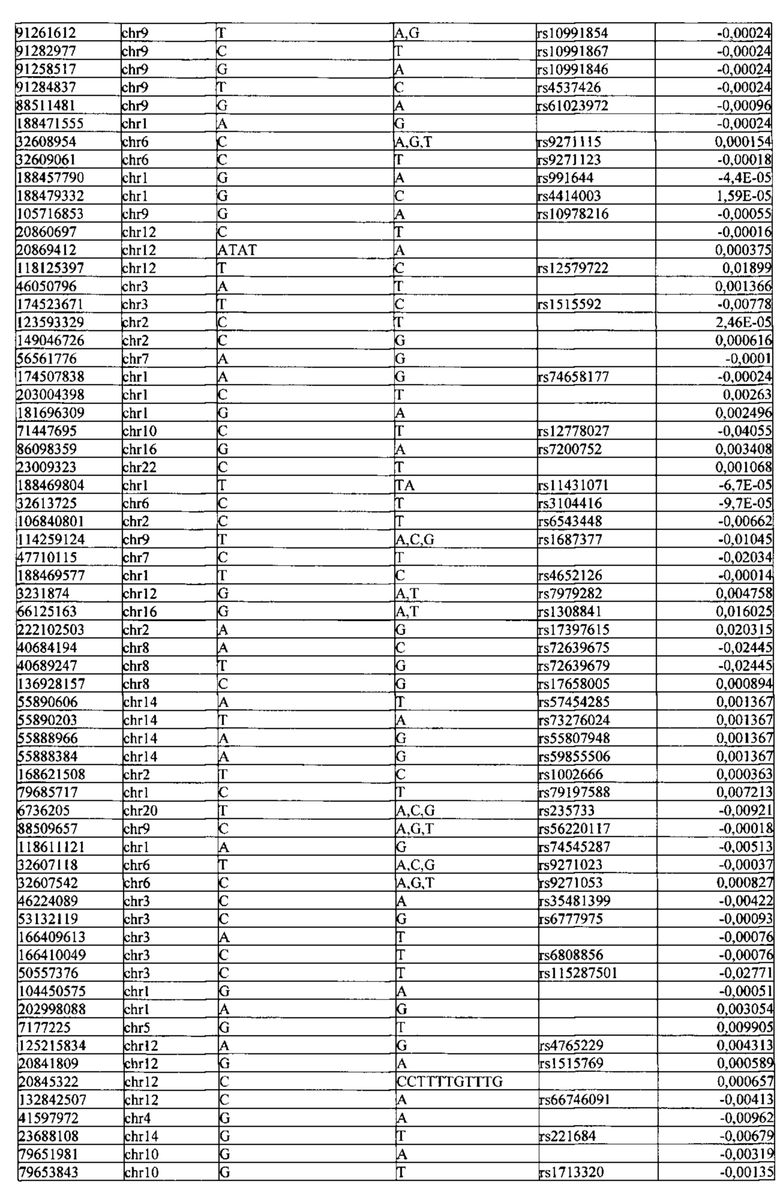
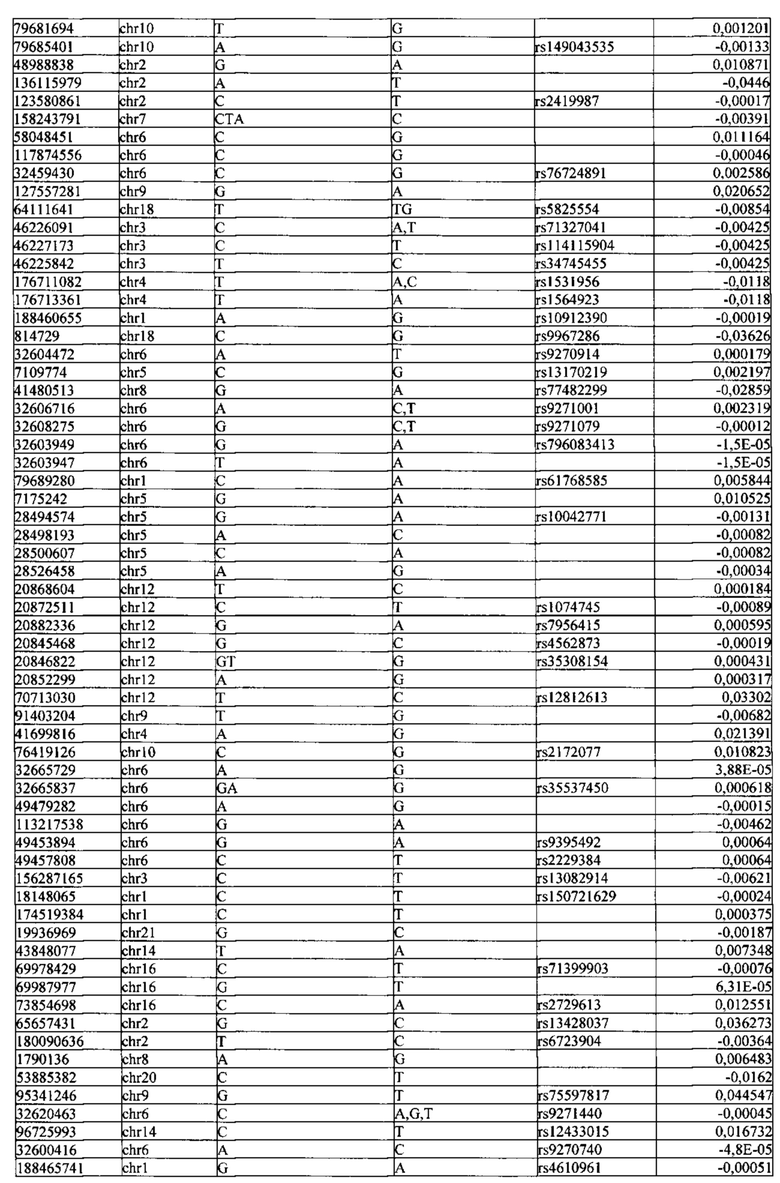
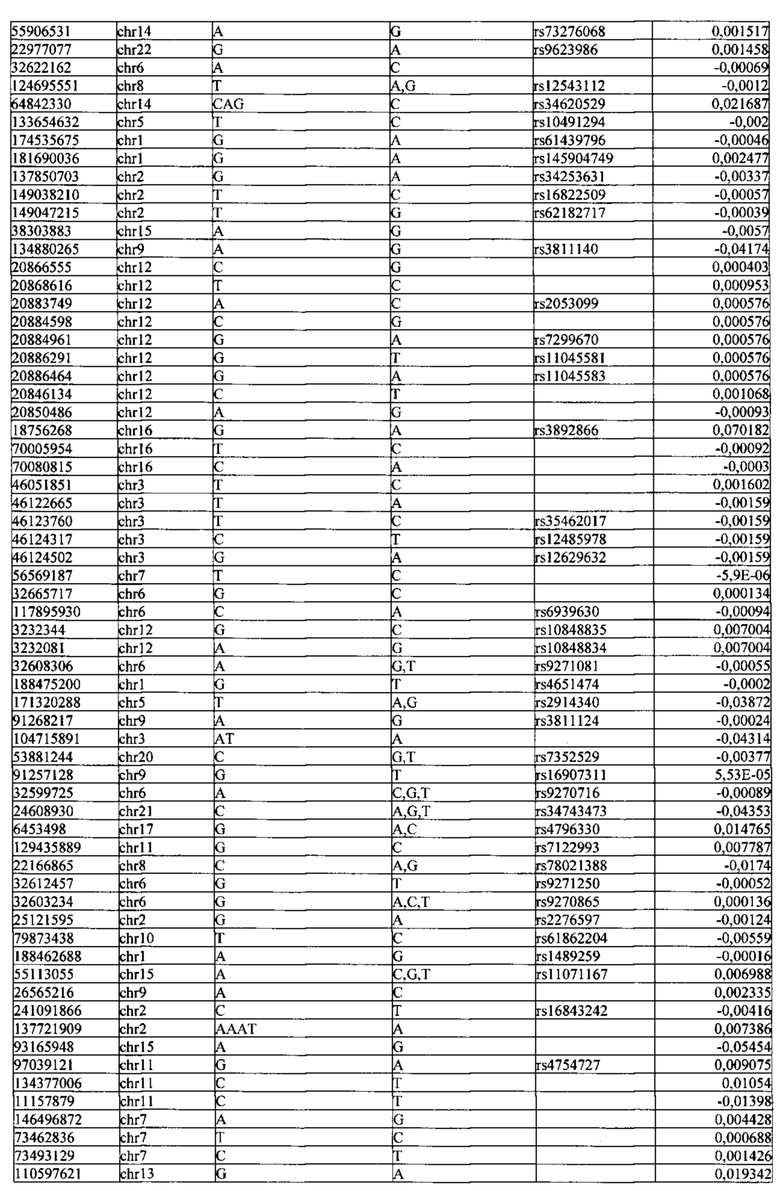
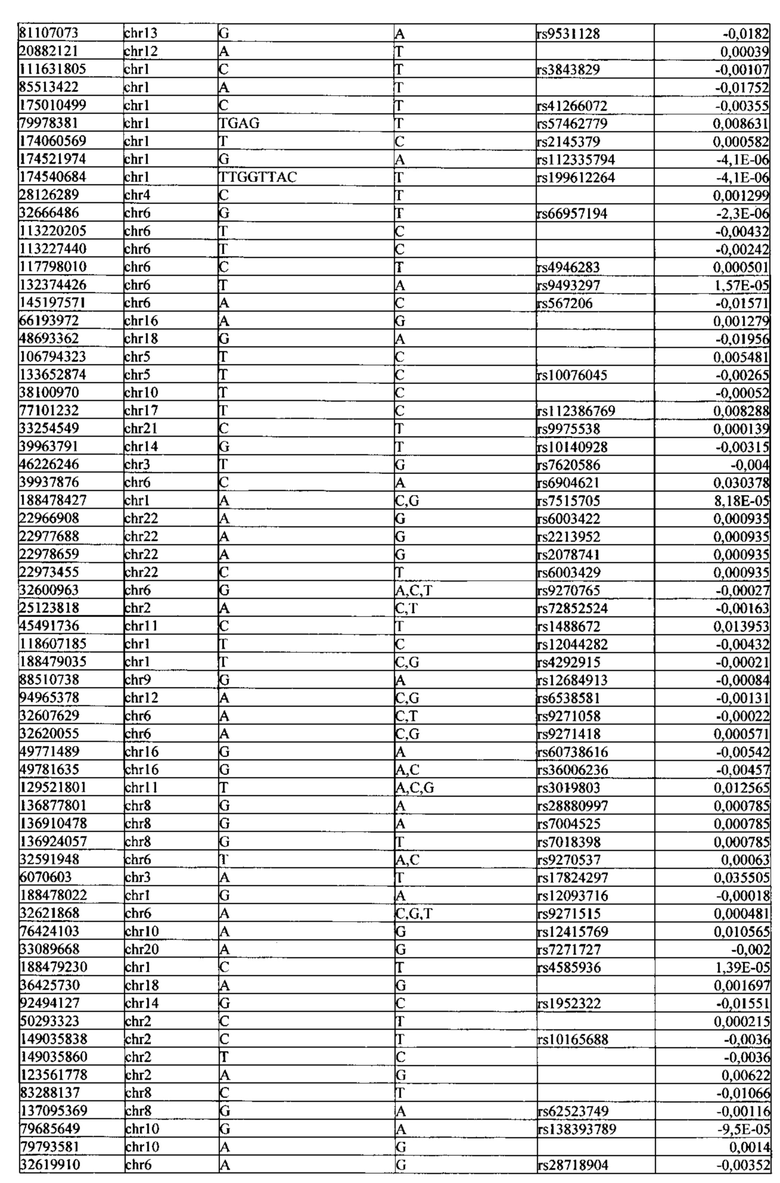
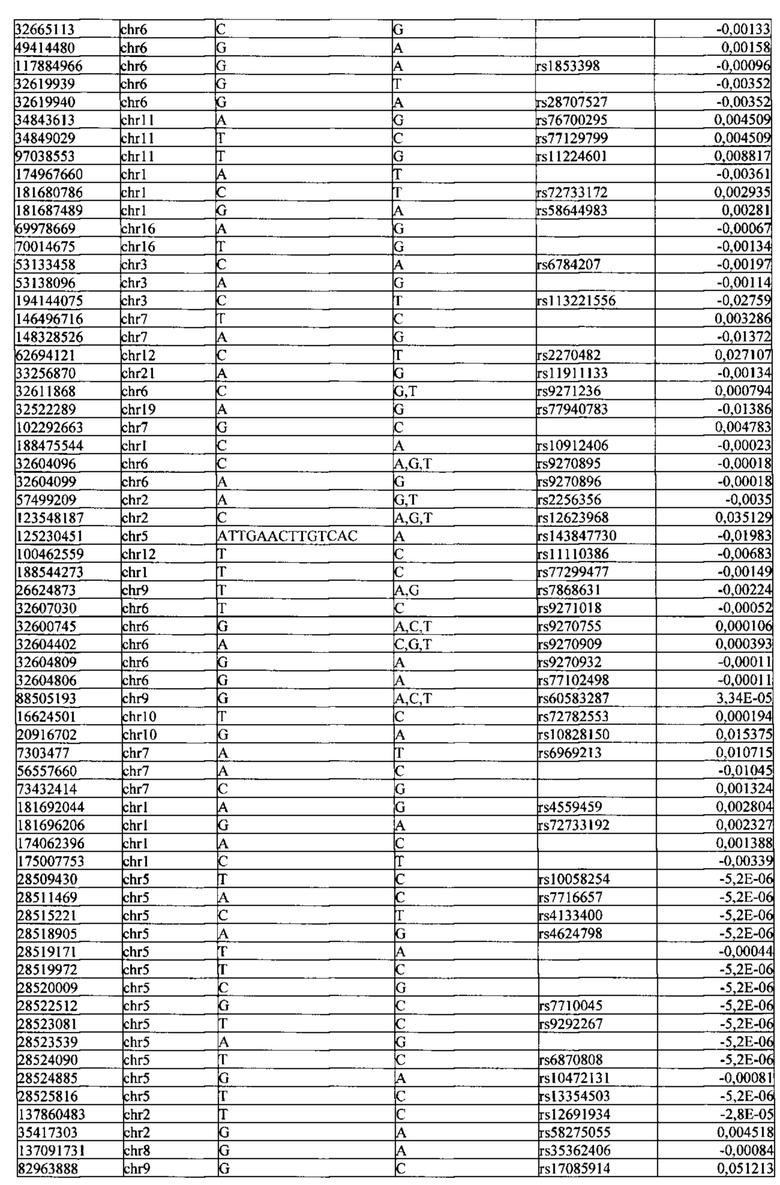
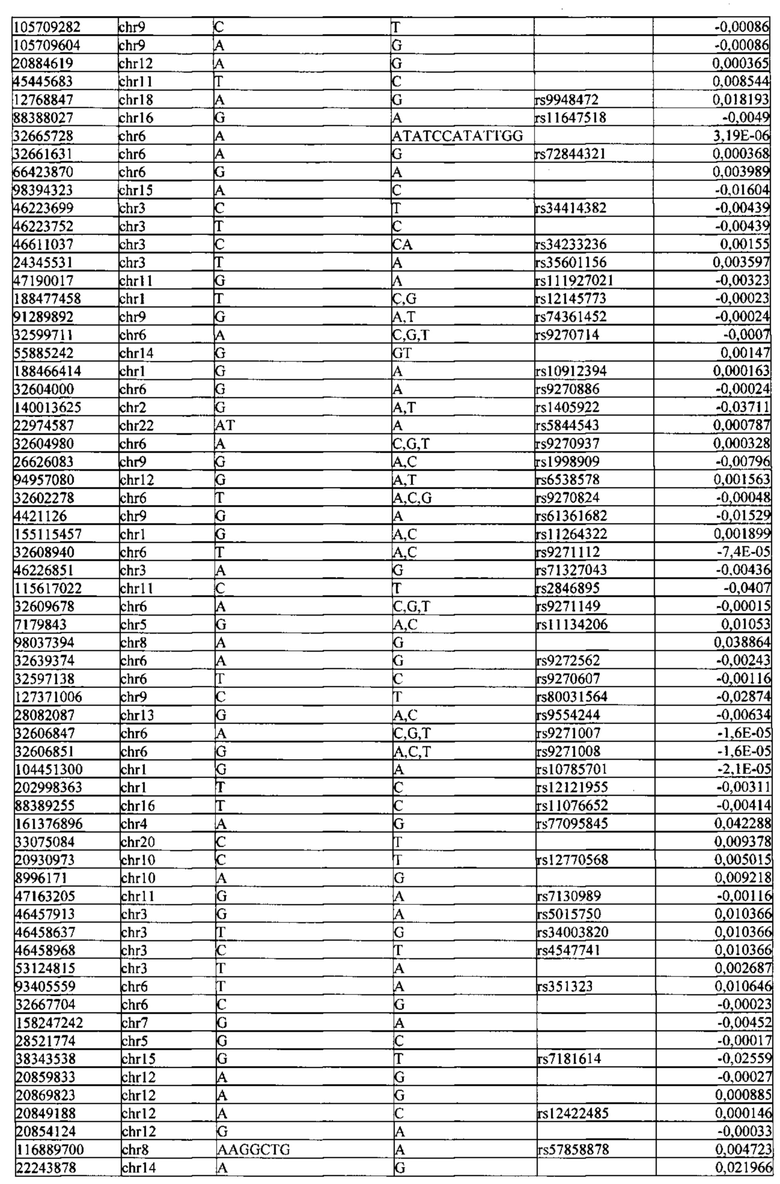
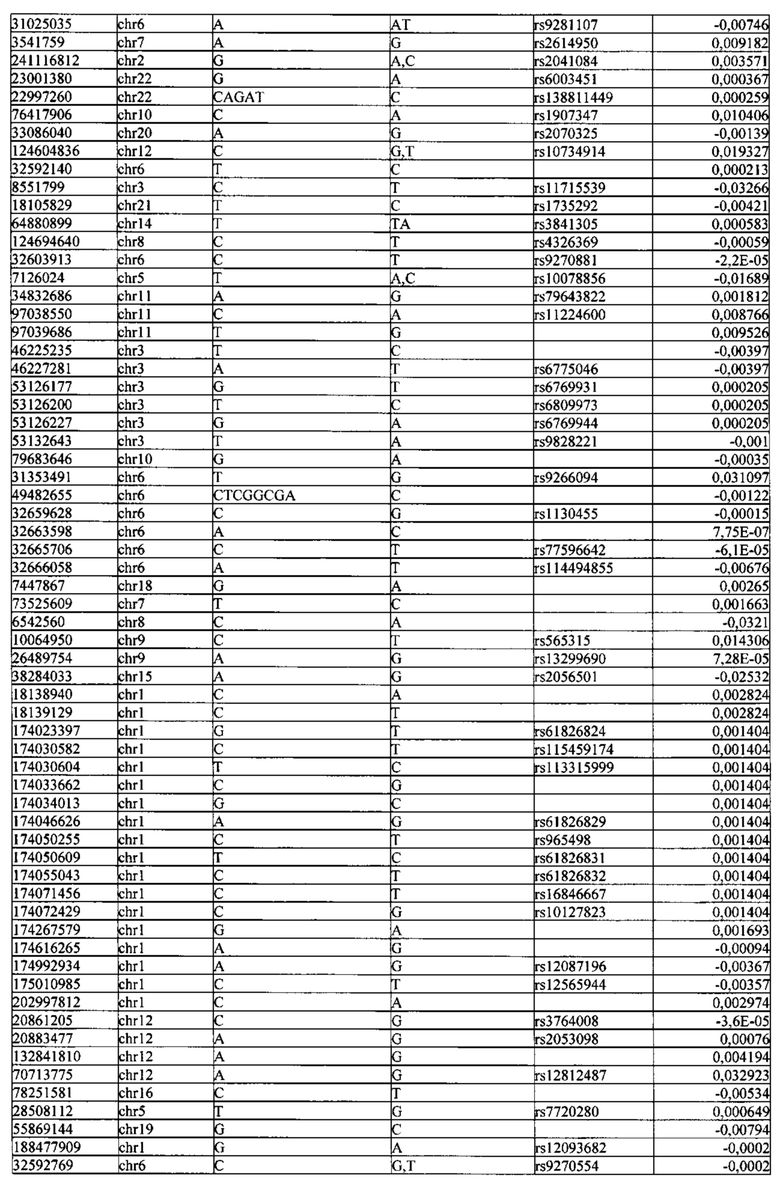
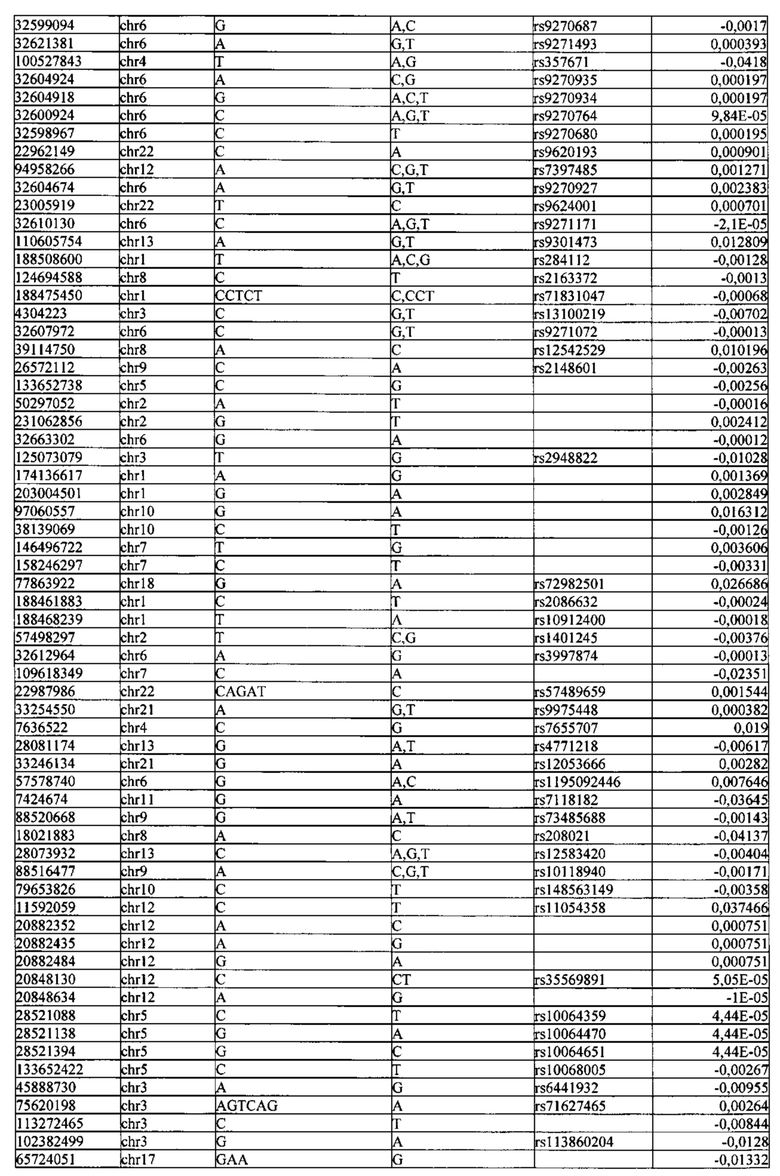
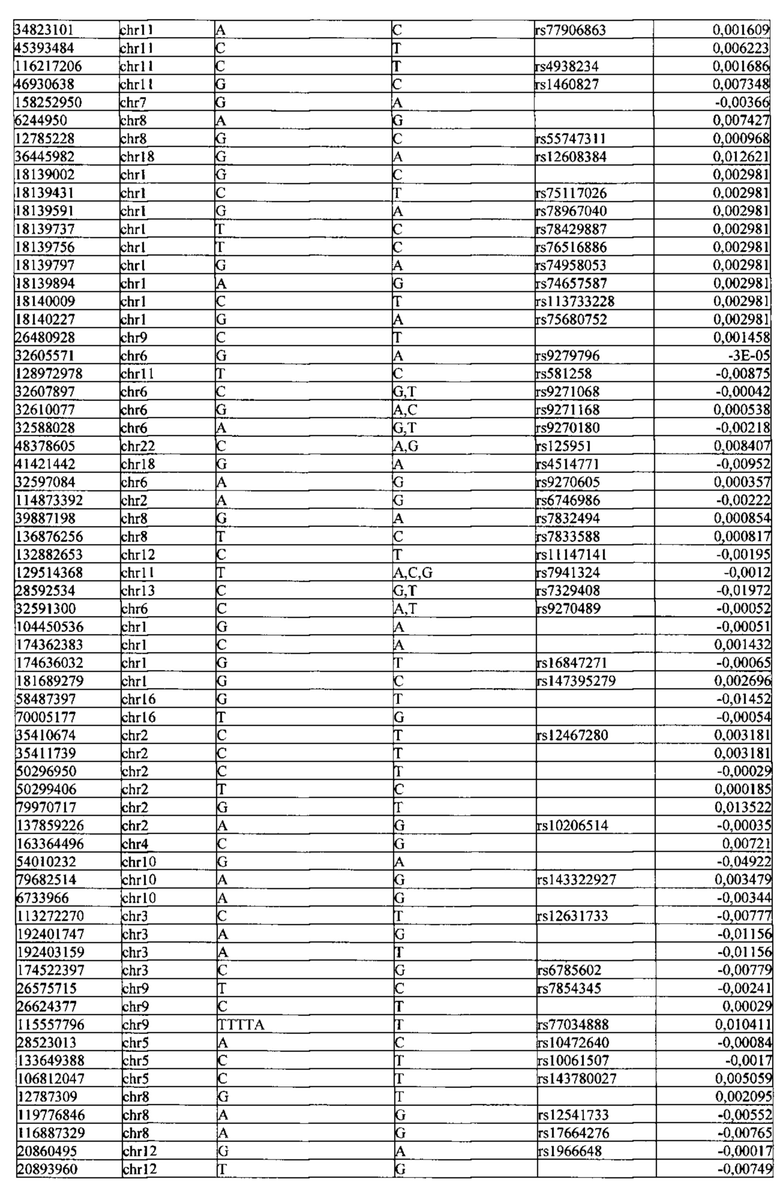
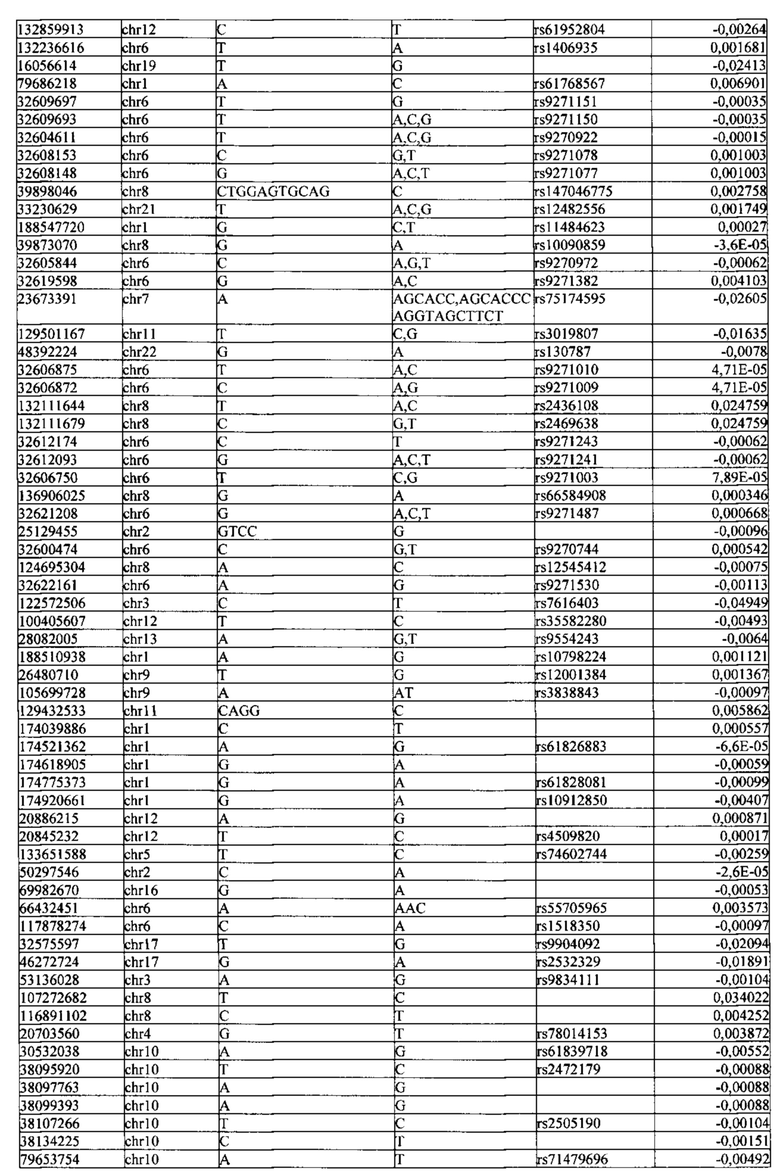
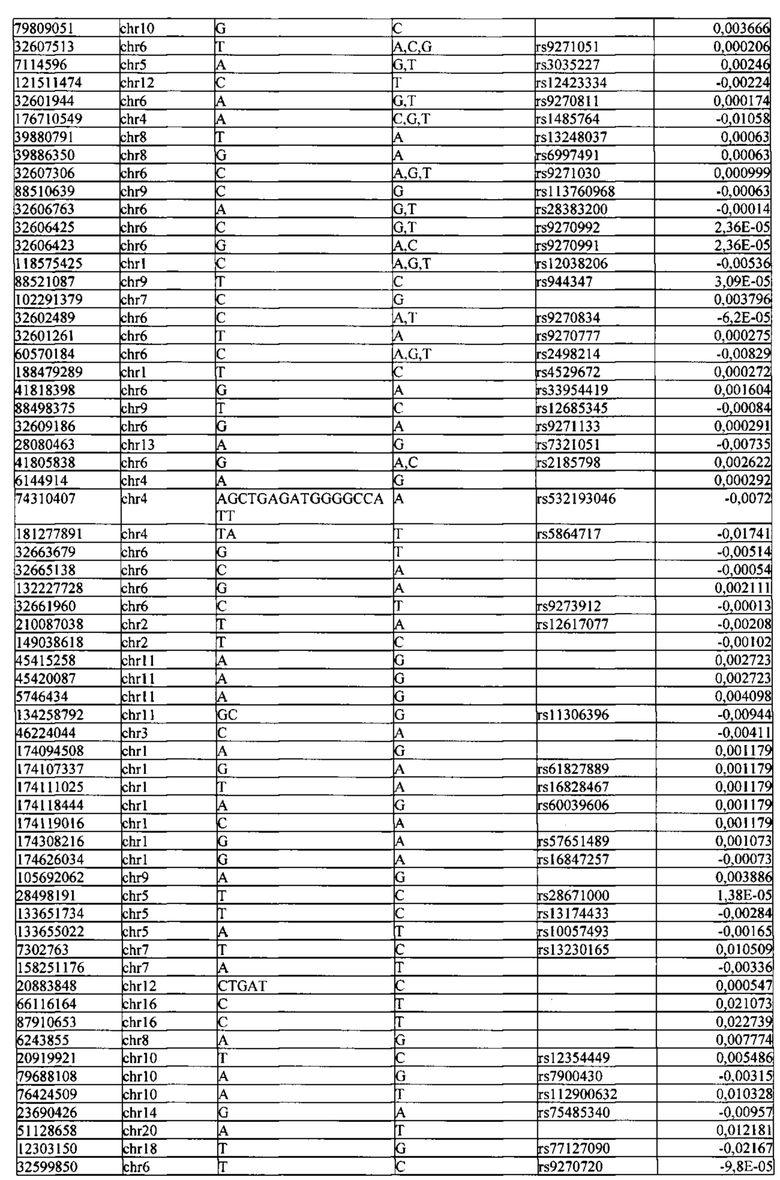
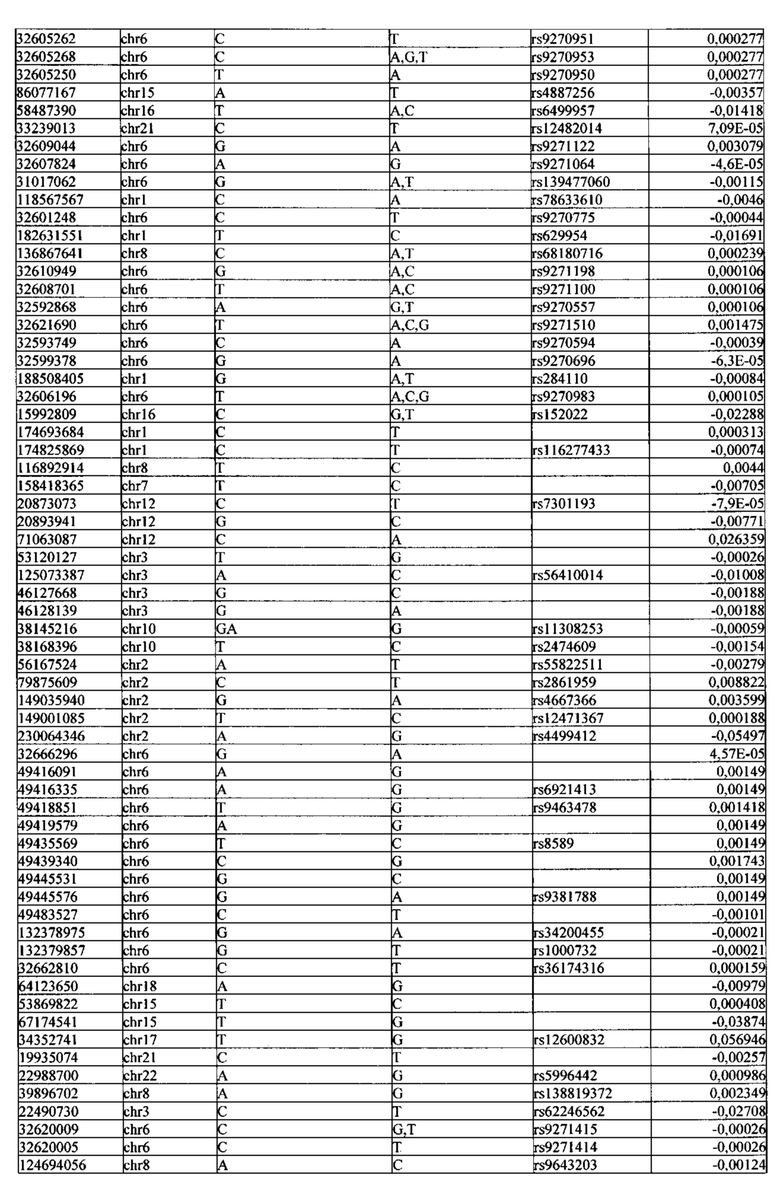
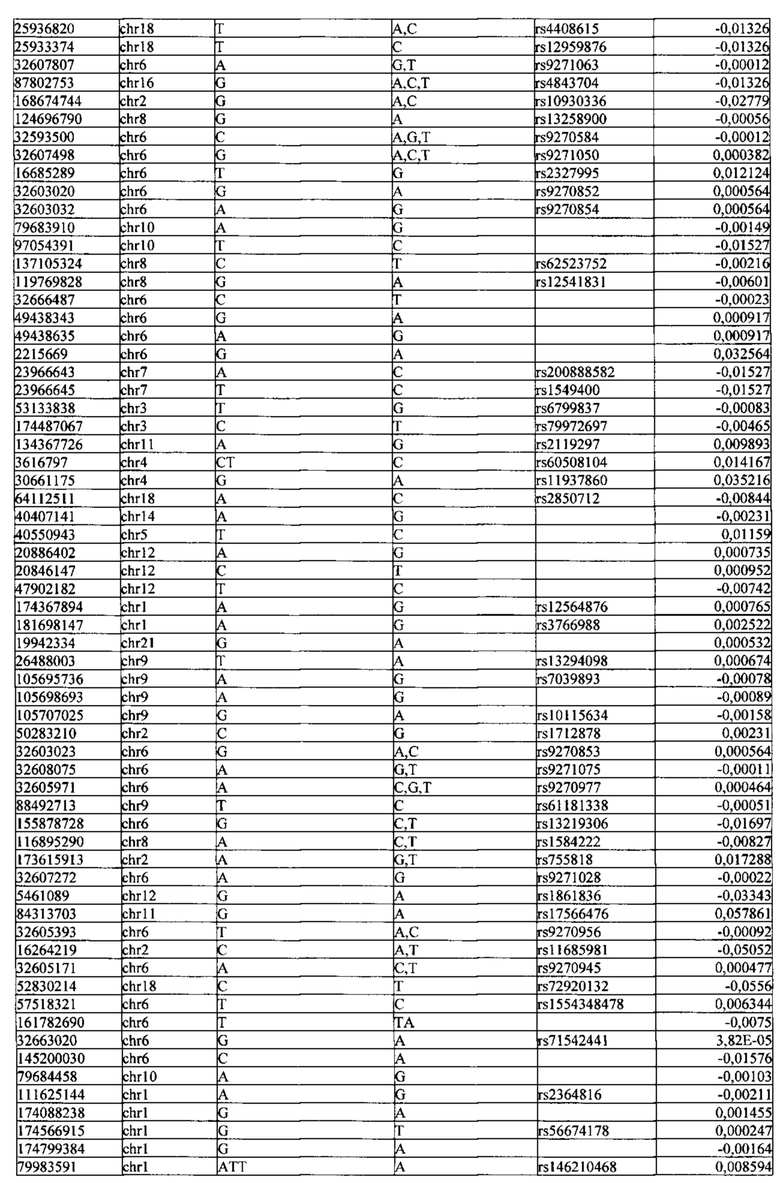
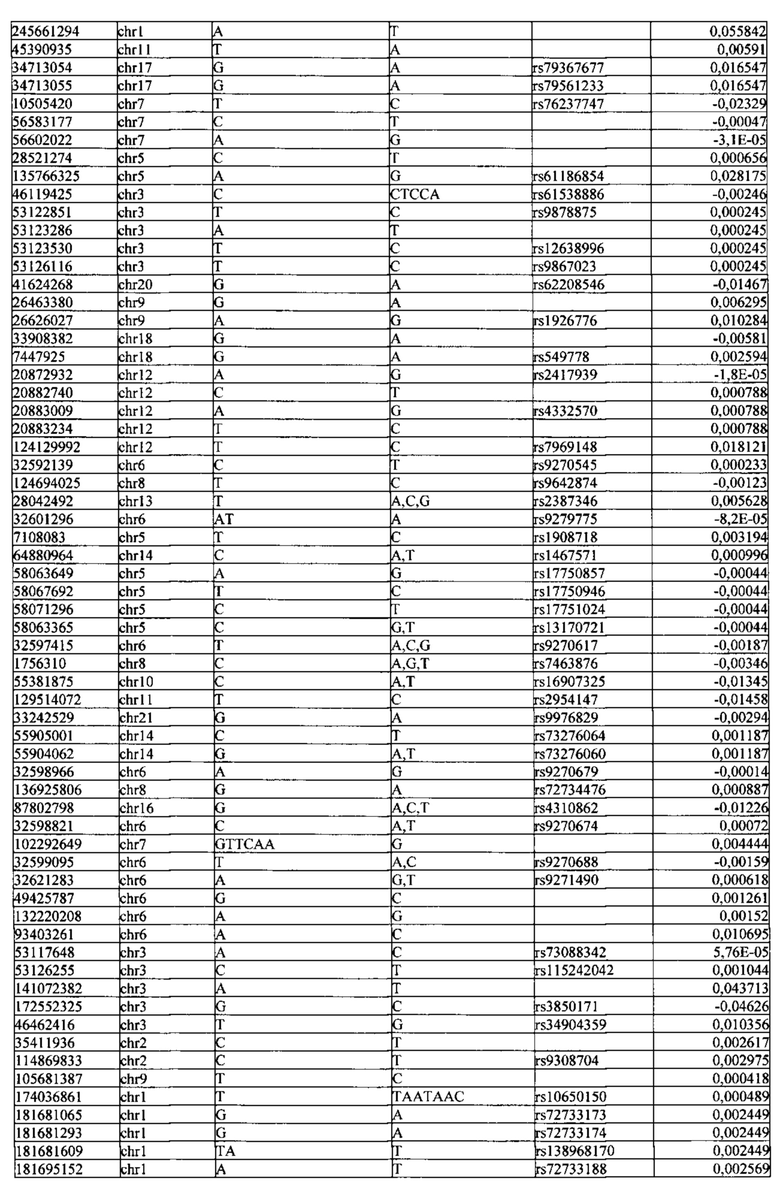
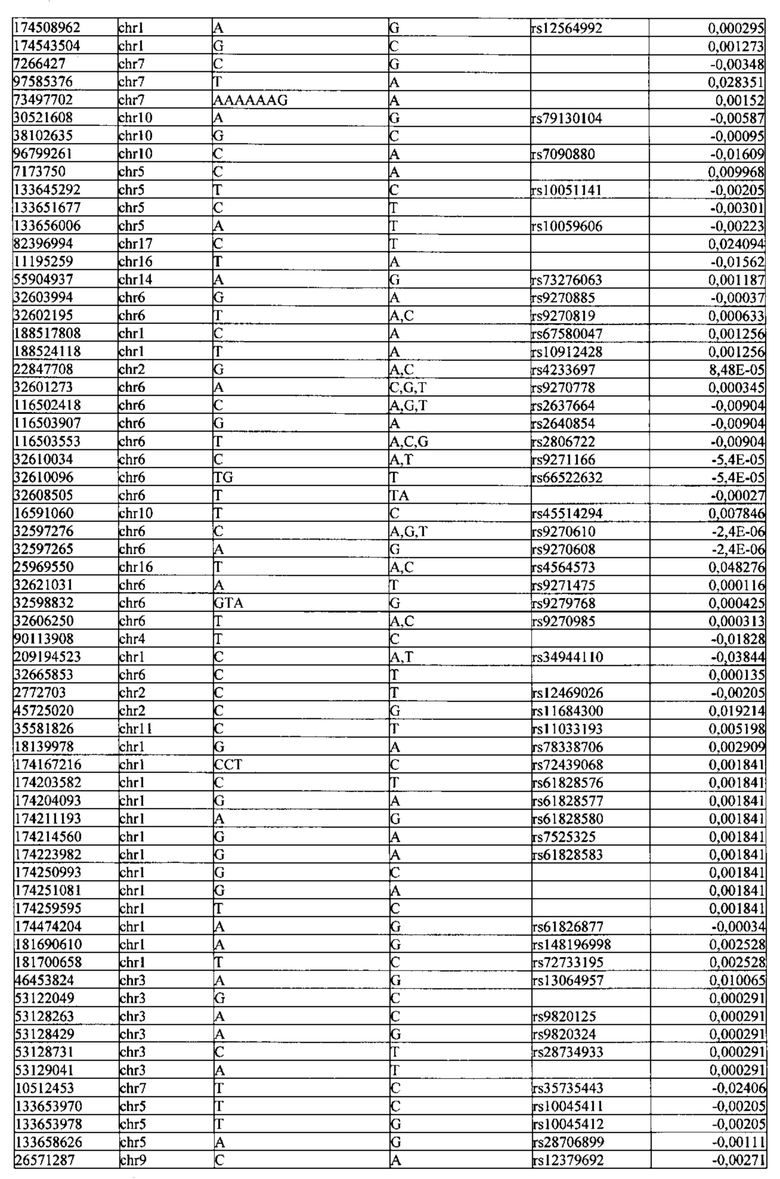
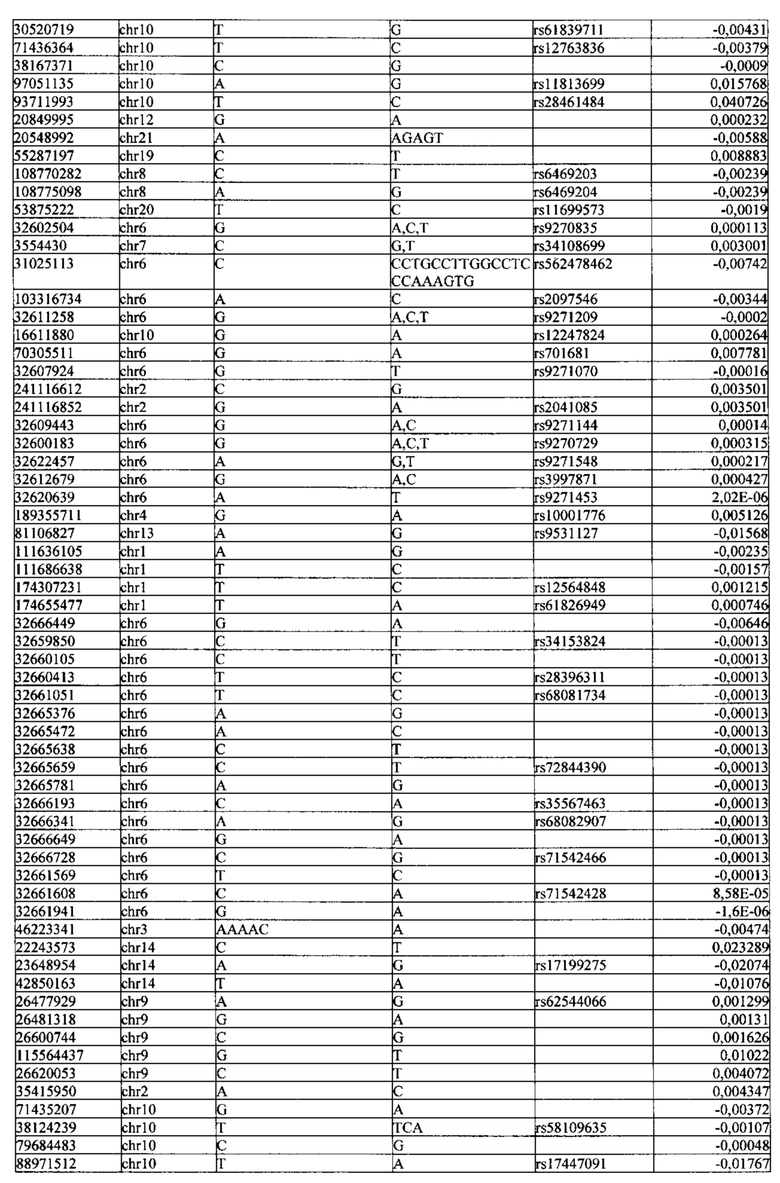
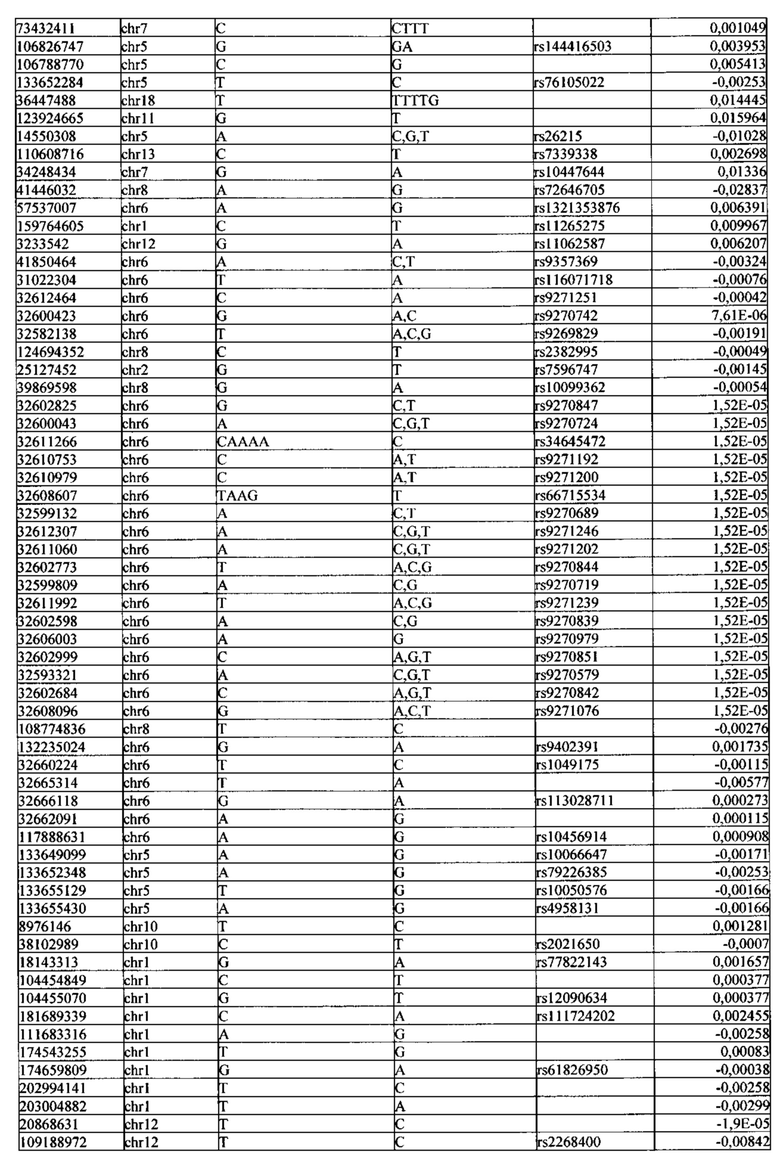
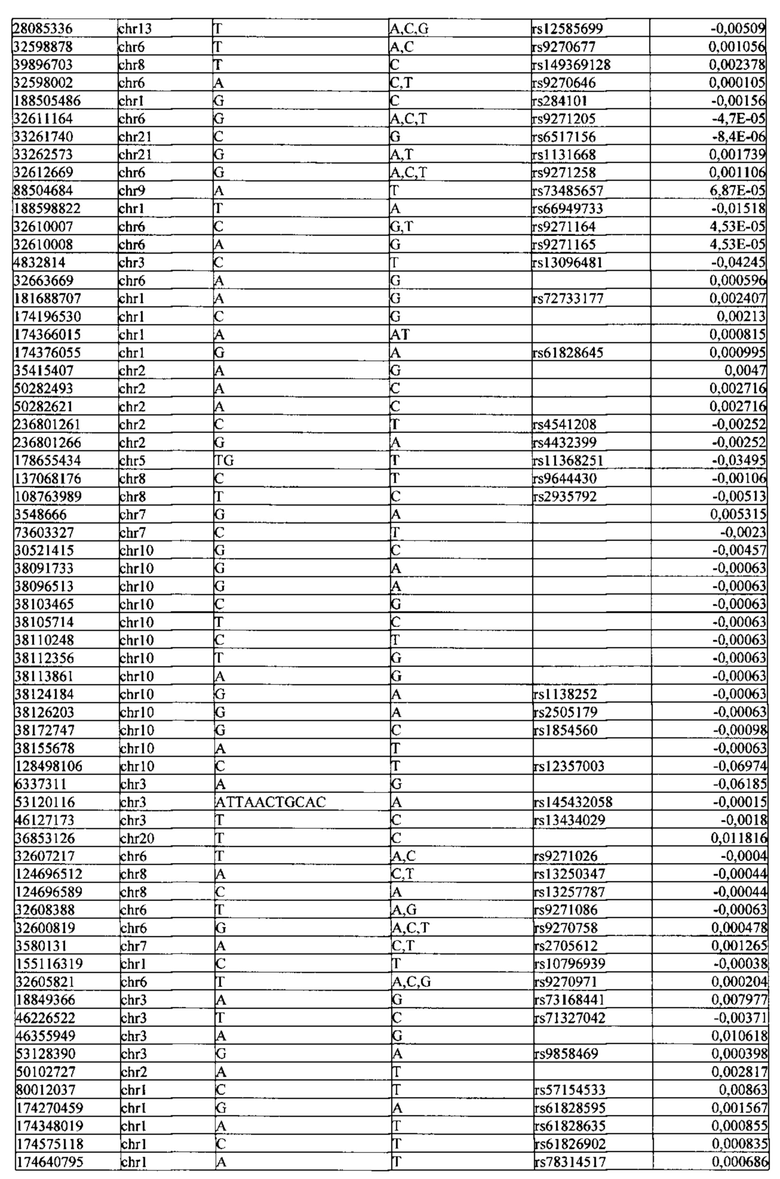
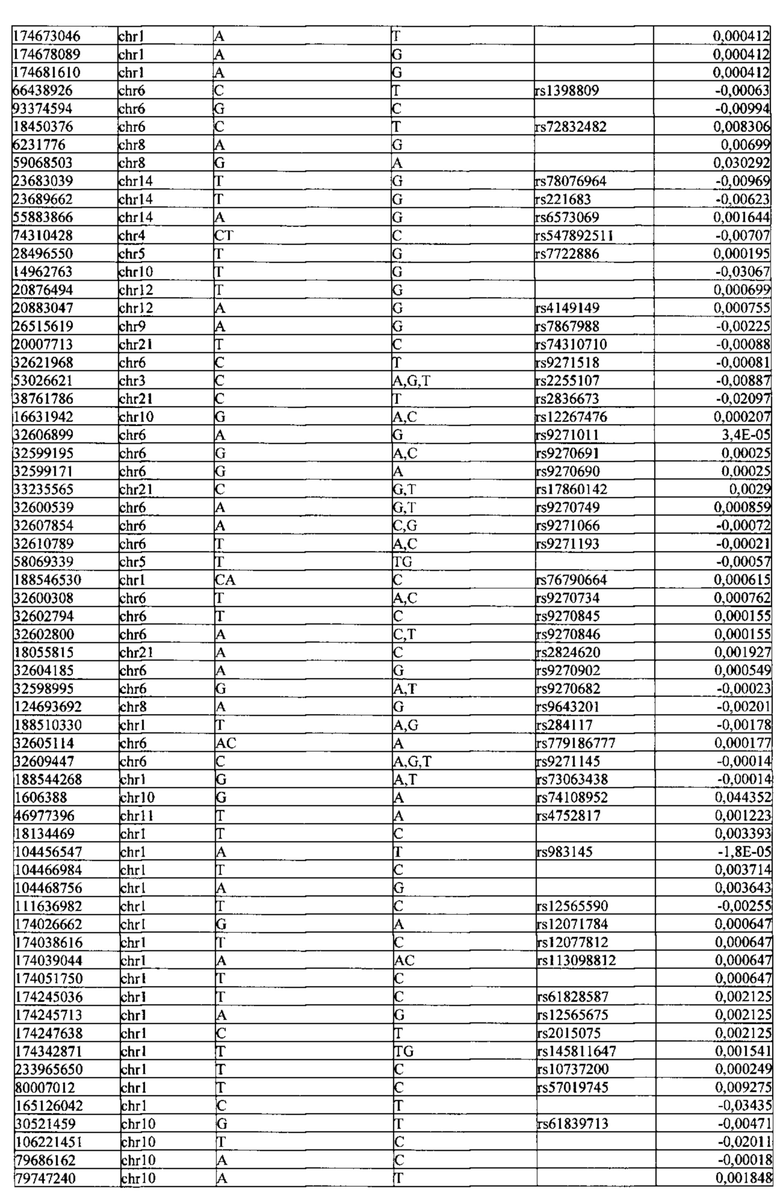
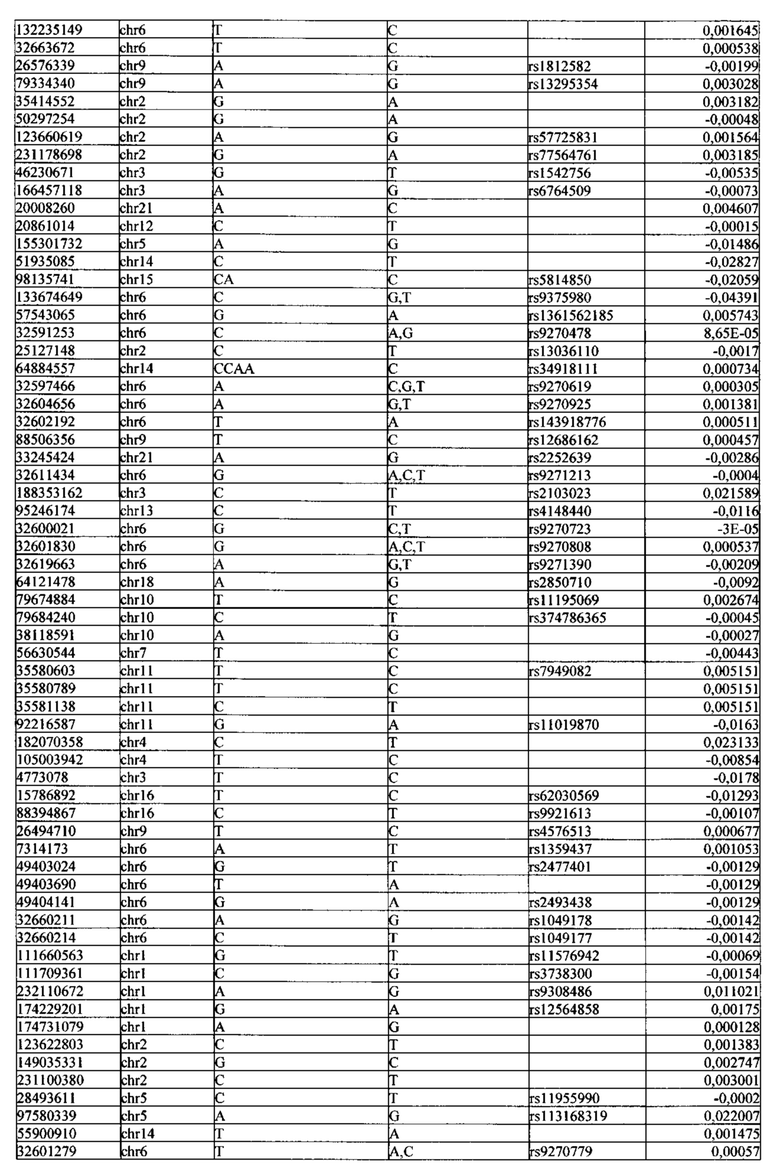
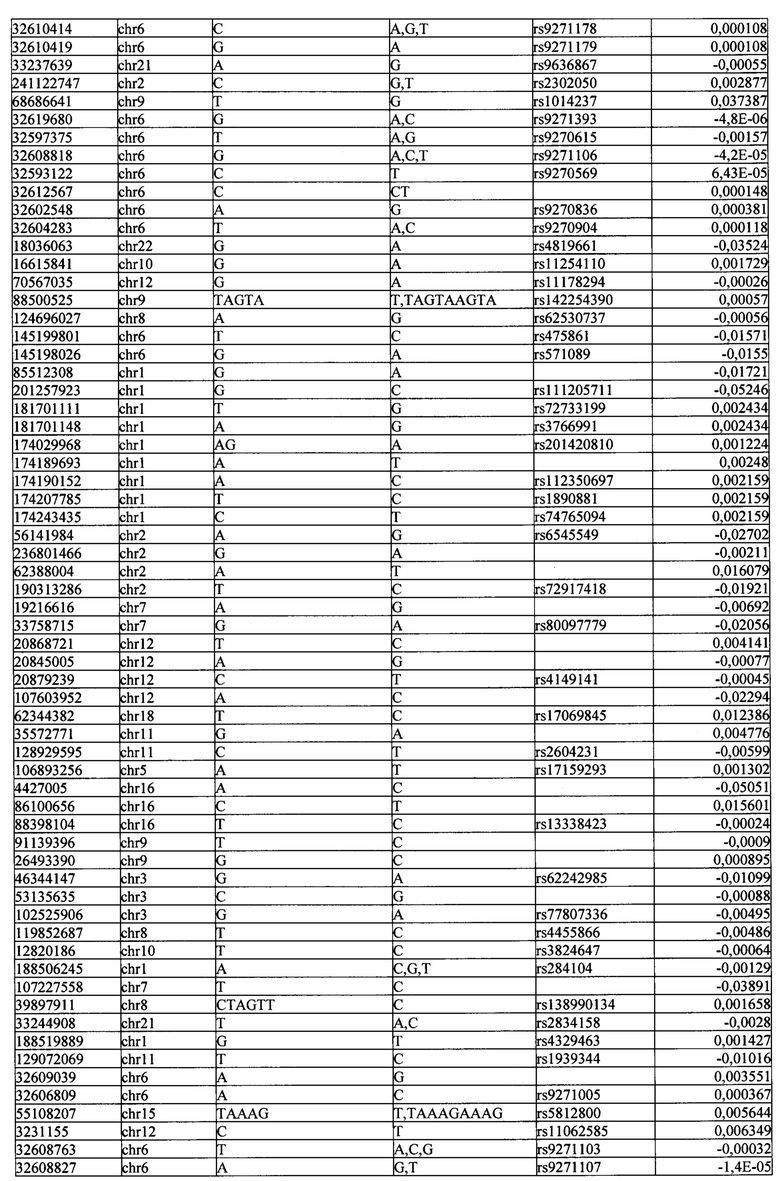
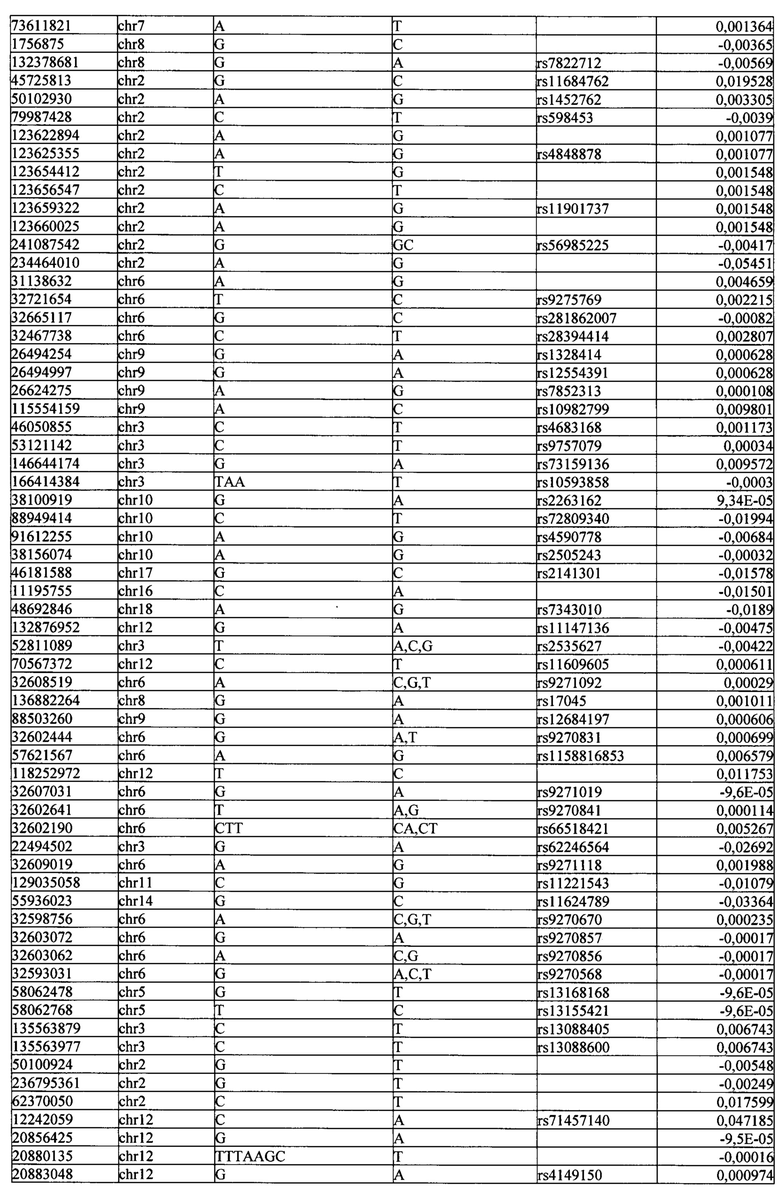
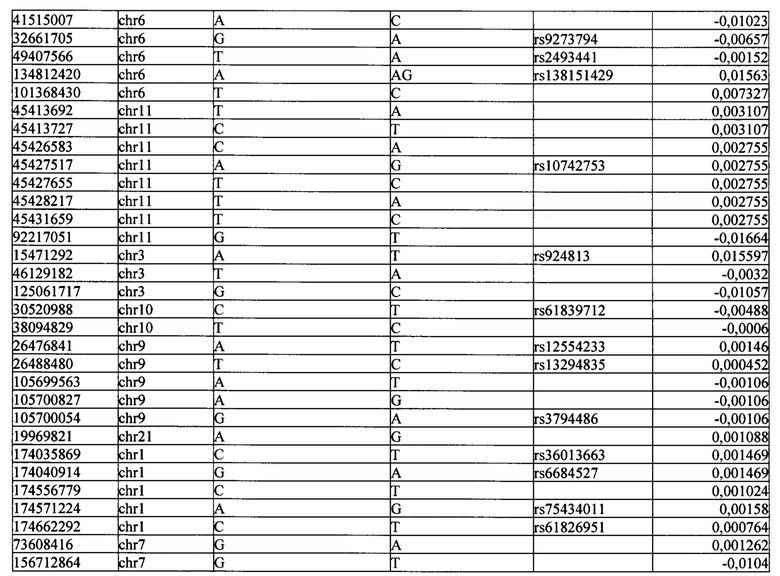
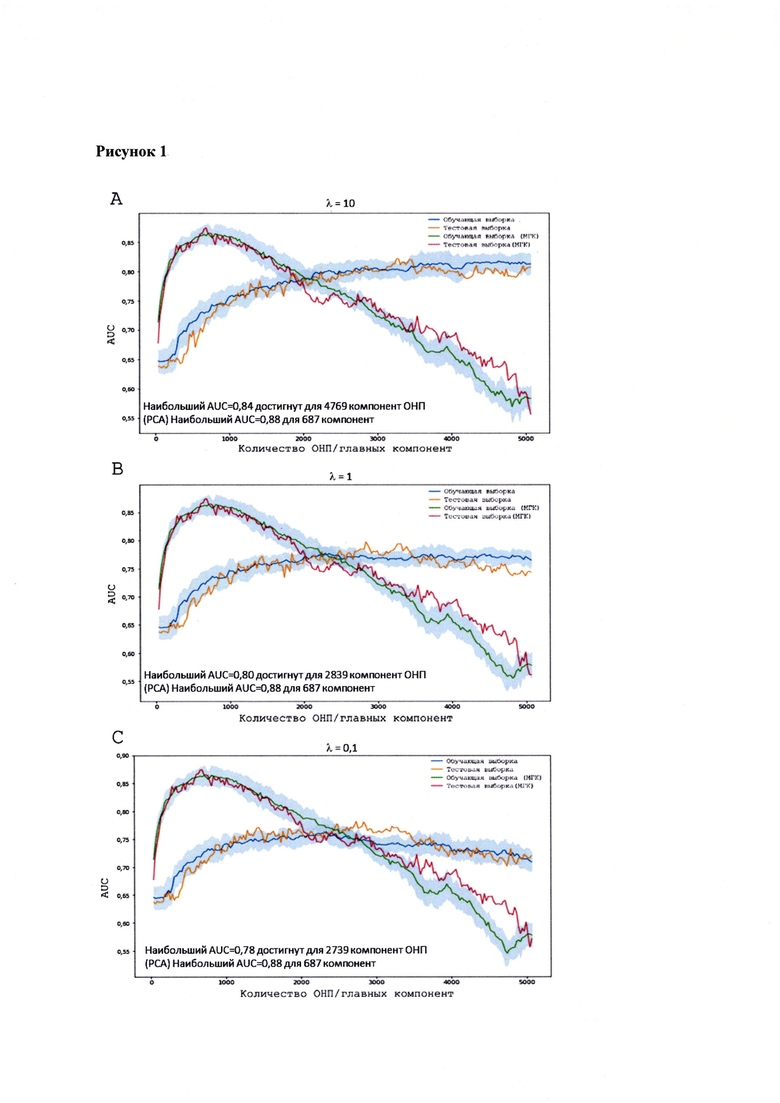
Поставленная задача решается тем, что в способе прогнозирования риска развития тяжелого течения COVID-19 у лиц на основании данных генетической диагностики методом секвенирования следующего поколения (NGS) выявляется факт наличия полиморфизмов, ассоциированных с тяжестью течения заболевания по результатам GWAS. Наиболее значимые из них расположенные на генах ZNF568, GPR173, PCDH15, IGSF3 (см. Таблица 2). Таблица 2 включает в себя позицию гена на хромосоме, номер хромосомы, нуклеотидную замену, идентификатор полиморфизма в виде rs кода, коэффициент обозначающий вес данного ОНП.
Предиктивная модель
Вероятность тяжелого течения заболевания вычисляется по формуле:
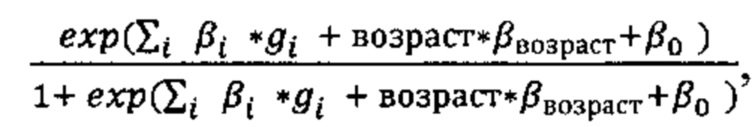 где
где 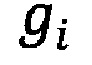 - количество аллелей риска, βi - коэффициент для данного полиморфизма, βвозраст - коэффициент, на который умножается возраст, β0 - свободный член (различается для мужчин и женщин); ехр - экспоненциальная функция. Выполнена валидация полигенной шкалы риска тяжелого течения COVID-19 на выборке из 5629 пациентов, в возрасте от 18 до 95 с известным клиническим течением COVID-19. Всем пациентам проводилось клиническое, лабораторное и инструментальное обследование на предмет исключения или подтверждения у них наличия SARS-CoV-2 инфекции, уточнения степени тяжести и генетическая диагностика.
- количество аллелей риска, βi - коэффициент для данного полиморфизма, βвозраст - коэффициент, на который умножается возраст, β0 - свободный член (различается для мужчин и женщин); ехр - экспоненциальная функция. Выполнена валидация полигенной шкалы риска тяжелого течения COVID-19 на выборке из 5629 пациентов, в возрасте от 18 до 95 с известным клиническим течением COVID-19. Всем пациентам проводилось клиническое, лабораторное и инструментальное обследование на предмет исключения или подтверждения у них наличия SARS-CoV-2 инфекции, уточнения степени тяжести и генетическая диагностика.
Критерии отбора больных для исследования.
Всем пациентам проводилось клиническое, лабораторное и инструментальное обследование на предмет исключения или подтверждения у них наличия SARS-CoV-2 инфекции, уточнения степени тяжести и генетическая диагностика. Пациенты были разделены на 2 группы: с легким течением заболевания (бессимптомное или легкое течение без вирусной пневмонии) и тяжелым течение заболевания (средне-тяжелое, тяжелое и крайне тяжелой течение заболевания). В основе деления на группы лежало наличие/отсутствие вирусной пневмонии.
Диагностику и оценку тяжести заболевания проводили в соответствии рекомендациями Минздрава РФ.
Бессимптомное течение
Легкое течение: температура тела 38°С, кашель, слабость, боли в горле; отсутствие критериев среднетяжелого и тяжелого течения;
Среднетяжелое течение: температура тела >38°С, ЧДД >22/мин, одышка при физических нагрузках; изменения при КТ (рентгенографии), типичные для вирусного поражения; SpO2<95%, СРБ сыворотки крови >10 мг/л;
Тяжелое течение: ЧДД>30/мин, SpO2<93%, PaO2 /FiO2≤300 мм рт. ст., Снижение уровня сознания, ажитация, нестабильная гемодинамика (систолическое АД менее 90 мм рт. ст. или диастолическое АД менее 60 мм рт. ст., диурез менее 20 мл/час); изменения в легких при КТ (рентгенографии), типичные для вирусного поражения (объем поражения значительный или субтотальный; КТ 3-4); лактат артериальной крови >2 ммоль/л; qSOFA>2 балла;
Крайне тяжелое течение: стойкая фебрильная лихорадка, ОРДС, ОДН с необходимостью респираторной поддержки (инвазивная вентиляции легких), септический шок, полиорганная недостаточность; изменения в легких при КТ (рентгенографии), типичные для вирусного поражения критической степени или картина ОРДС.
Подобное разделение было обусловлено стремлением провести оценку воздействия генетических факторов, установленных в результате полногеномного поиска ассоциаций, на развитие пневмонии и серьезных осложнений при COVID-19. Бессимптомные участники и участники со слабо выраженной симптоматикой не страдали пневмонией. По данным КТ-диагностики у пациентов с умеренной, тяжелой и крайне тяжелой симптоматикой была диагностирована вирусная пневмония (см. Таблица 1).
Генетическая диагностика
Кровь от пациентов получена кубитальным венозным доступом. При заборе крови исследователи руководствуются ГОСТ Р 53079.4-2008. Для получения образцов цельной крови кровь забирается с помощью закрытых систем взятия крови S-Monovette Sarstedt (К2-ЭДТА) с фиолетовой крышкой. Хранение крови проводилось при температуре -20°С.
Выделение геномной ДНК из образцов цельной крови проводилось с использованием автоматизированной станции Tecan Freedom EVO (Tecan, Швейцария) при помощи набора MagAttract HMW DNA Kit (Qiagen, Германия), а также применялась процедура ручного выделения с применением набора DNA Blood Mini Kit (Qiagen, Германия) в соответствии с протоколом производителя.
Концентрация и чистота выделенной ДНК определялась двумя методами: в автоматическом режиме на планшетном ридере Infinite® 200 Pro с помощью программы Magellan™, а также в ручном режиме - концентрация ДНК измерялась на флуориметре Qubit 4.0 (Thermo Fisher Scientific, США), оценка качества производилась на NanoDropTM One С Microvolume UV-Vis (Thermo Fisher Scientific, США).
Для всех образцов выделенной ДНК отношение показателей поглощения 260/280 составило 1,8-2,0, отношение 260/230 - 2,0-2,2.
Для приготовления библиотек использовали 150-500 нг геномной ДНК. Тагментацию ДНК, очистку и амплификацию тагментированнной ДНК, очистку полученных библиотек проводили согласно протоколу Nextera DNA Flex Library Prep (Document # 1000000025416 v07, Illumina, США). Полногеномные библиотеки готовили с использованием наборареагентов Nextera DNA Flex kit (Illumina, США), согласно рекомендациям производителя, и набора индексов IDT-ILMN Nextera DNA UD Indexes Set А и Set В для предотвращения кросс-контаминации образцов. Концентрация библиотек измерялась на планшетном ридере Infinite® F Nano Plus. Размер полученных библиотек определяли при помощи набора реагентов Agilent D1000 на приборе Agilent 4200 TapeStation (Agilent Technologies, США). Пулирование производилось автоматически с использованием роботизированной станции Tecan Freedom EVO (Tecan, Швейцария). Пул библиотек, содержащий 24 образца, перед секвенированием разводили до концентрации 2,8 нМ. Контроль качества пулов проводили при помощи набора реагентов Agilent HS D1000 ScreenTape на приборе Agilent 4200 TapeStation (Agilent Technologies, США). Полногеномное секвенирование проводили на приборе Illumina NovaSeq 6000, используя набор реагентов S4, 300 циклов (Illumina, США) с парно-концевыми прочтениями 2×150 п. о.
Выравнивание на референсный геном (GRCh38) проводили на платформе «Dragen Bio-IT» (производства компании «Illumina», США); определение вариантов проводили на определителе Strelka2 для исследований небольших когорт {фильтрация по качеству}. Варианты, не соответствующие критерию равновесия Харди-Вайнберга (р<10е-6), мультиаллельные варианты и варианты с частотой минорного аллеля менее 1% были исключены из исследования.
Статистический анализ
Для проверки полногеномных ассоциаций проводили логистическую регрессию:
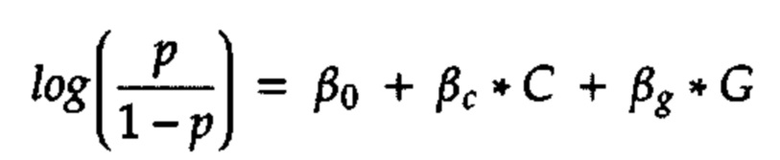
где, β0 - постоянная величина, βс - коэффициент для вектора ковариант, С - вектор ковариат, βg - коэффициент для вектора генотипов, G - вектор генотипов. Оптимизация проводилась для каждой позиции, прошедшей процедуру фильтрации. Целевой показатель (тяжесть протекания заболевания) был зашифрован в виде двоичной переменной (для тяжелых случаев заболевания использовали код 1). Расчеты проводились с использованием библиотеки «Python» («statsmodels v0.12.2»), распараллеленной на «Spark Cluster.» Возраст, пол и 10 первых координат компонент учитывали как коварианты. Во второй вариант GWAS в качестве коварианта был добавлен ИМТ. После фильтрации по качеству было протестировано 7 944 406 вариантов. Для прогнозирования риска была построена модель с гребневой регрессией (Python v3.8.). Коэффициенты регрессии находятся при помощи минимизации суммы квадратов разности наблюдаемых и полученных значений и ее регуляризация методом добавления "штрафа" в виде суммы квадратов значений коэффициентов:
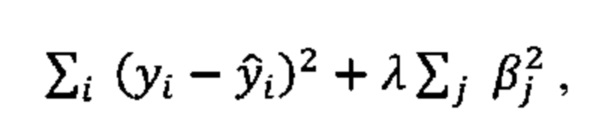
у - наблюдаемые значения, 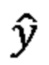 - полученные значения, β - коэффициенты моделью, λ - параметр регуляризации. ОНП для модели выбирали на основе их показателя по результатам GWAS, расчет которого проводился следующим образом:
- полученные значения, β - коэффициенты моделью, λ - параметр регуляризации. ОНП для модели выбирали на основе их показателя по результатам GWAS, расчет которого проводился следующим образом: 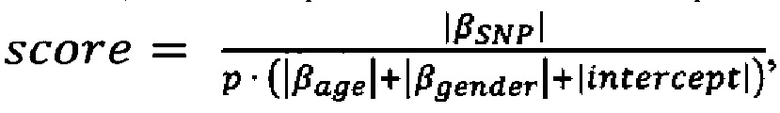 βSNP - коэффициент регрессии для полиморфизма, βage - коэффициент регрессии для возрастной переменной, βgender - коэффициент регрессии для тендерной переменной, intercept - свободный член, р - р-значение для ОНП. Рассматривались только ОНП с частотой >0,15. Далее проводился анализ главных компонент. Для выбора оптимального количества ОНП/главных компонент была построена модель для каждого варианта от 1 до размера выборки. Каждую модель проверяли методом перекрестной (десятикратной) проверки и тестировали на 10% образцов. Путем максимизации показателя AUC (area under the curve или площадь под кривой) для тестового набора данных методом последовательных приближений выбирали несколько значений параметра регуляризации λ; λ=0,1, 1,10.
βSNP - коэффициент регрессии для полиморфизма, βage - коэффициент регрессии для возрастной переменной, βgender - коэффициент регрессии для тендерной переменной, intercept - свободный член, р - р-значение для ОНП. Рассматривались только ОНП с частотой >0,15. Далее проводился анализ главных компонент. Для выбора оптимального количества ОНП/главных компонент была построена модель для каждого варианта от 1 до размера выборки. Каждую модель проверяли методом перекрестной (десятикратной) проверки и тестировали на 10% образцов. Путем максимизации показателя AUC (area under the curve или площадь под кривой) для тестового набора данных методом последовательных приближений выбирали несколько значений параметра регуляризации λ; λ=0,1, 1,10.
Полученные результаты
Для прогнозирования риска тяжелого течения COVID-19 мы построили полигенную шкалу риска на основе 689 компонент, полученных в результате анализа 5121 ОНП с самыми высокими показателями. Процесс выбора показан на Рисунке 1. Изменение параметра регуляризации не влиял на точность модели при заданных компонентах. Поэтому было выбрано значение λ=1, позволяющее наиболее быстро провести вычисления. Значение AUC конечной модели составило 0,95 (Рис. 2). Коэффициенты ОНП приведенные в Таблице 2 были получены обратным переходом от пространства главных компонент к исходному.
Пример 1. Мужчина, 41 год, с легким течение COVID-19 (заболевание сопровождалось аносмией, дизгевзией, слабостью, повышением температуры тела более 37°С), получал амбулаторное лечение. В результате полногеномного секвенирования образцов крови данного пациента было обнаружено 3130 аллели риска среди рассматриваемых. Их суммирование с коэффициентами из таблицы 2 (Приложение) дает значение 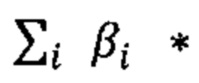
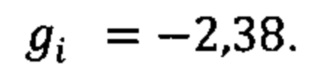 Прибавим к этой величине возраст умноженный на коэффициент из Таблицы 2 βвозраст=0,02 и константу β0=0,71 для мужчин. Получаем значение в скобках для формулы
Прибавим к этой величине возраст умноженный на коэффициент из Таблицы 2 βвозраст=0,02 и константу β0=0,71 для мужчин. Получаем значение в скобках для формулы 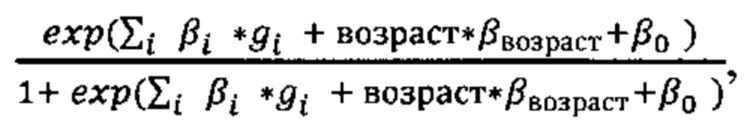 -2,38+41*0,02+0,71=-0,75. Рассчитываем риск тяжелого течения COVID-19
-2,38+41*0,02+0,71=-0,75. Рассчитываем риск тяжелого течения COVID-19 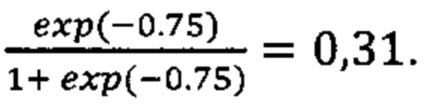 Значение меньше 0,5, следовательно предполагается легкое течение заболевания, что было подтверждено анамнезом.
Значение меньше 0,5, следовательно предполагается легкое течение заболевания, что было подтверждено анамнезом.
Пример 2. Женщина, 39 года, с тяжелым течением COVID-19 (у пациентки имелась вирусная пневмония с дыхательной недостаточностью). В результате полногеномного секвенирования образцов крови данного пациента было обнаружено 4563 аллели риска среди рассматриваемых. Их суммирование с коэффициентами из таблицы 2 (Приложение) дает значение 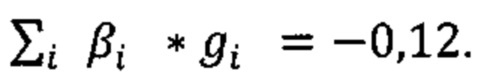 Прибавим к этой величине возраст умноженный на коэффициент из Таблицы 2 βвозраст=0,02 и константу β0=0,48 для женщин (из таблицы 2). Получаем значение в скобках для формулы
Прибавим к этой величине возраст умноженный на коэффициент из Таблицы 2 βвозраст=0,02 и константу β0=0,48 для женщин (из таблицы 2). Получаем значение в скобках для формулы 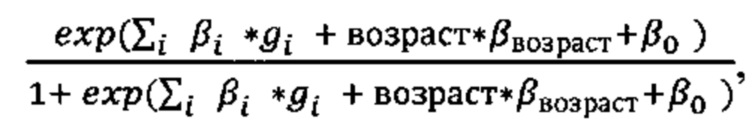 -0,12+39*0,02+0,48=1,24. Рассчитываем риск тяжелого течения COVID-19
-0,12+39*0,02+0,48=1,24. Рассчитываем риск тяжелого течения COVID-19 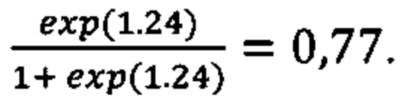 Значение больше 0,5, следовательно предполагается тяжелое течение заболевания, что было подтверждено анамнезом.
Значение больше 0,5, следовательно предполагается тяжелое течение заболевания, что было подтверждено анамнезом.
Перечень литературы:
1. Huang, Chaolin, Yeming Wang, Xingwang Li, Lili Ren, Jianping Zhao, Yi Hu, Li Zhang, et al. 2020. "Clinical Features of Patients Infected with 2019 Novel Coronavirus in Wuhan, China." The Lancet 395 (10223): 497-506.
2. Chen, Nanshan, Min Zhou, Xuan Dong, Jieming Qu, Fengyun Gong, Yang Han, Yang Qiu, et al. 2020. "Epidemiological and Clinical Characteristics of 99 Cases of 2019 Novel Coronavirus Pneumonia in Wuhan, China: A Descriptive Study." The Lancet 395 (10223): 507-13.
3. Coronaviridae Study Group of the International Committee on Taxonomy of Viruses. 2020. "The Species Severe Acute Respiratory Syndrome-Related Coronavirus: Classifying 2019-nCoV and Naming It SARS-CoV-2." Nature Microbiology 5 (4): 536-44.
4. Aiewsakun, Pakorn, Patrawee Nilplub, Patompon Wongtrakoongate, Suradej Hongeng, and Arunee Thitithanyanont. 2021. "SARS-CoV-2 Genetic Variations Associated with COVID-19 Pathogenicity." Microbial Genomics 7 (12). https://doi.org/10.1099/mgen.0.000734.
5. "Russian Federation: WHO Coronavirus Disease (COVID-19) Dashboard with Vaccination Data." n.d. Accessed February 8, 2022. https://covidl9.who.int/region/euro/country/ru.
6. Ellinghaus, D., F. Degenhardt, L. Bujanda, M. Buti, and Others. 2020. "Genomewide Association Study of Severe Covid-19 with Respiratory Failure. NEJM."
7. Li, Yuanfeng, Yuehua Ke, Xinyi Xia, Yahui Wang, Fanjun Cheng, Xinyi Liu, Xin Jin, et al. 2021. "Genome-Wide Association Study of COVID-19 Severity among the Chinese Population." Cell Discovery 7 (1): 76.
8. Pairo-Castineira, Erola, Sara Clohisey, Lucija Klaric, Andrew D. Bretherick, Konrad Rawlik, Dorota Pasko, Susan Walker, et al. 2021. "Genetic Mechanisms of Critical Illness in COVID-19." Nature 591 (7848): 92-98.
9. Yun, Hyunok, Nan lee Noh, and Eun Young Lee. 2022. "Genetic Risk Scores Used in Cardiovascular Disease Prediction Models: A Systematic Review." Reviews in Cardiovascular Medicine 23 (1): 8.
10. Forrest, Iain S., Kumardeep Chaudhary, Ishan Paranjpe, Ha My T. Vy, Carla Marquez-Luna, Ghislain Rocheleau, Aparna Saha, et al. 2021. "Genome-Wide Polygenic Risk Score for Retinopathy of Type 2 Diabetes." Human Molecular Genetics 30 (10): 952-60.
11. Prakrithi, P., Priya Lakra, Durai Sundar, Manav Kapoor, Mitali Mukerji, Ishaan Gupta, and The Indian Genome Variation Consortium. 2021. "Genetic Risk Prediction of COVID-19 Susceptibility and Severity in the Indian Population." Frontiers in Genetics 12 (October), https://doi.org/10.3389/fgene.2021.714185.
Приложение
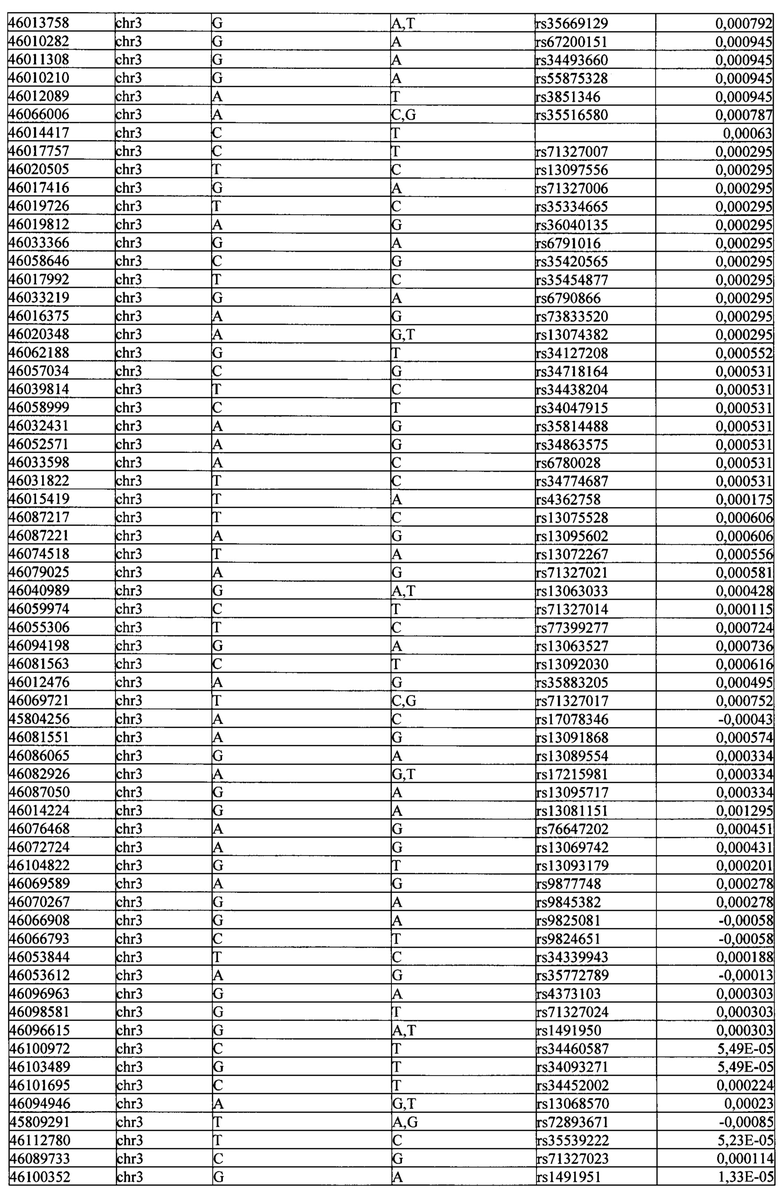
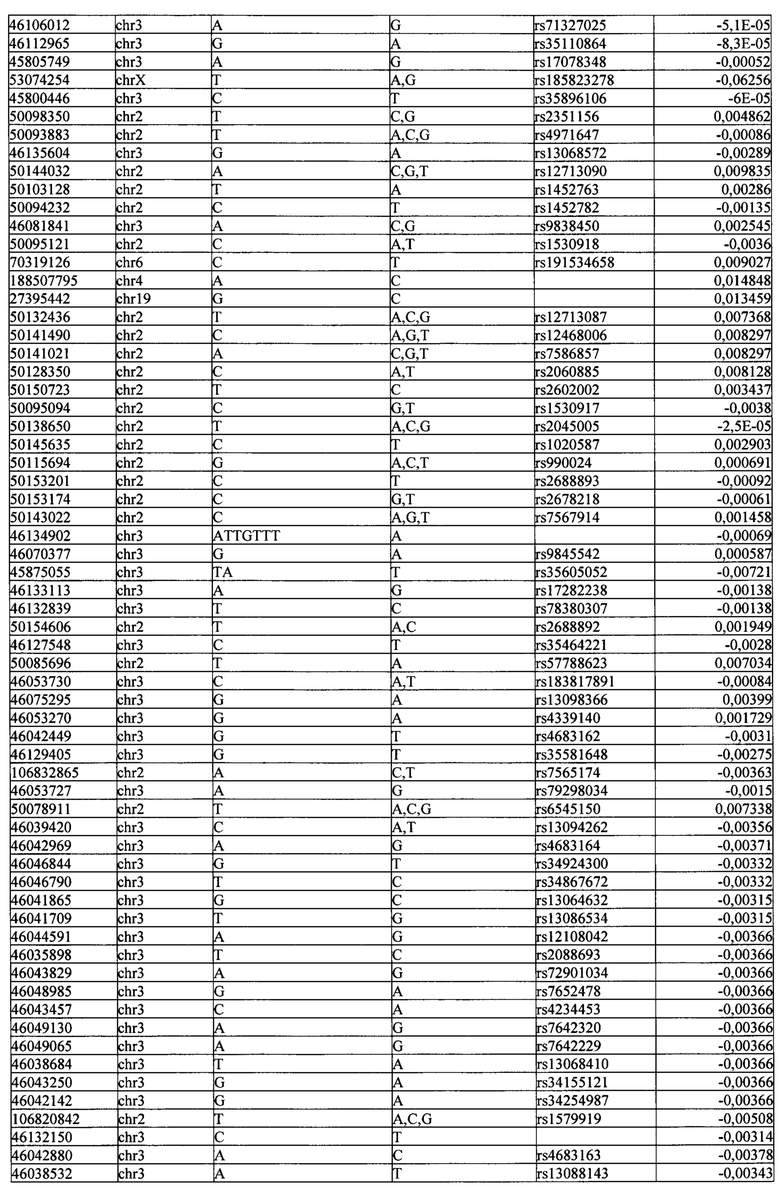
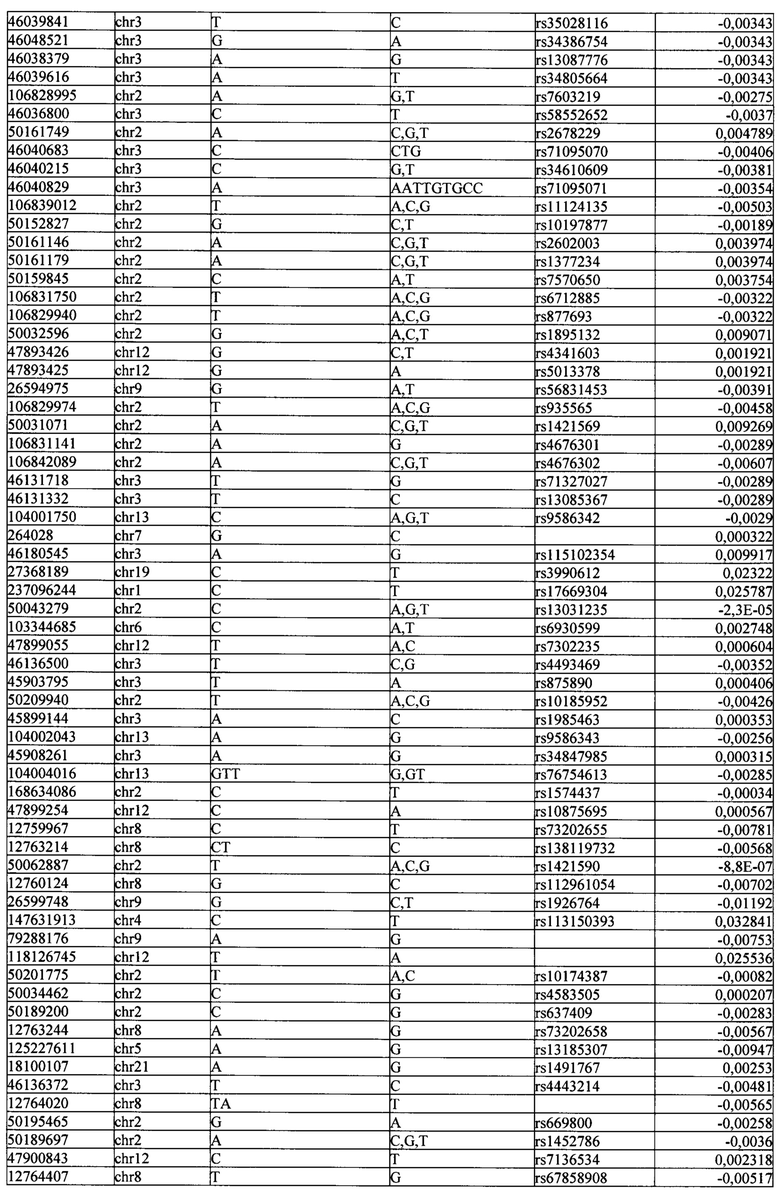
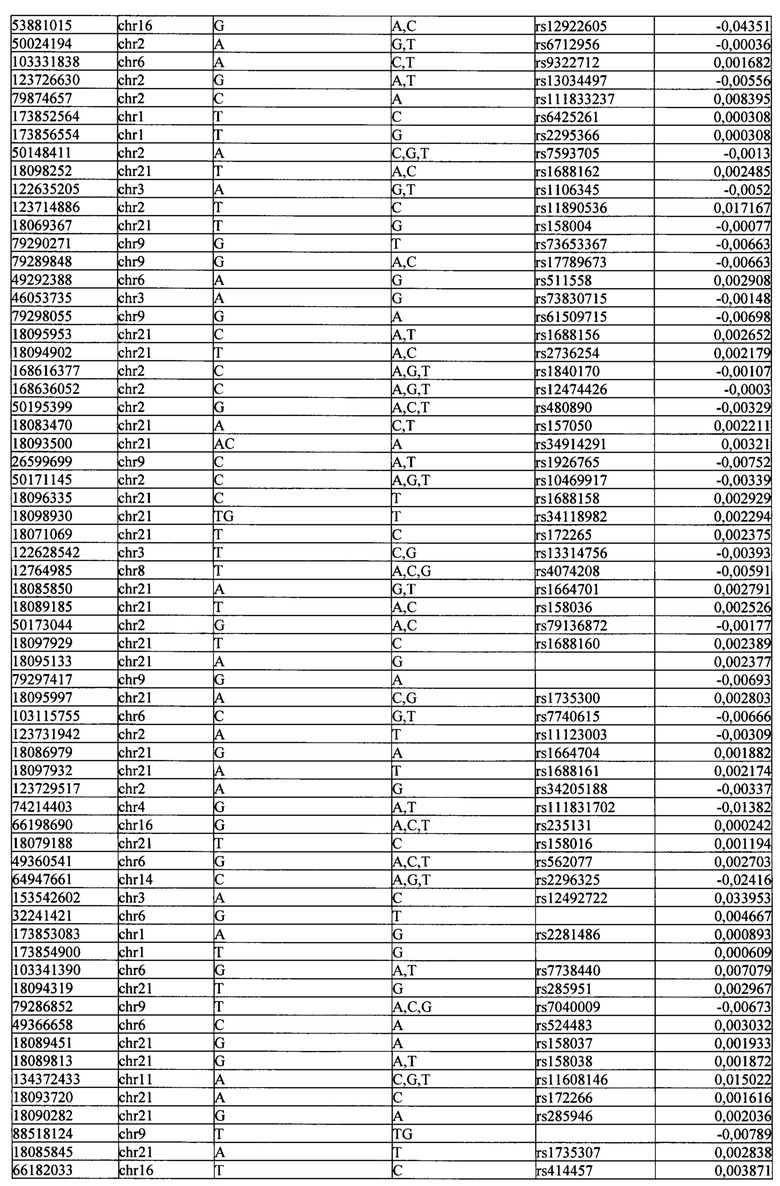
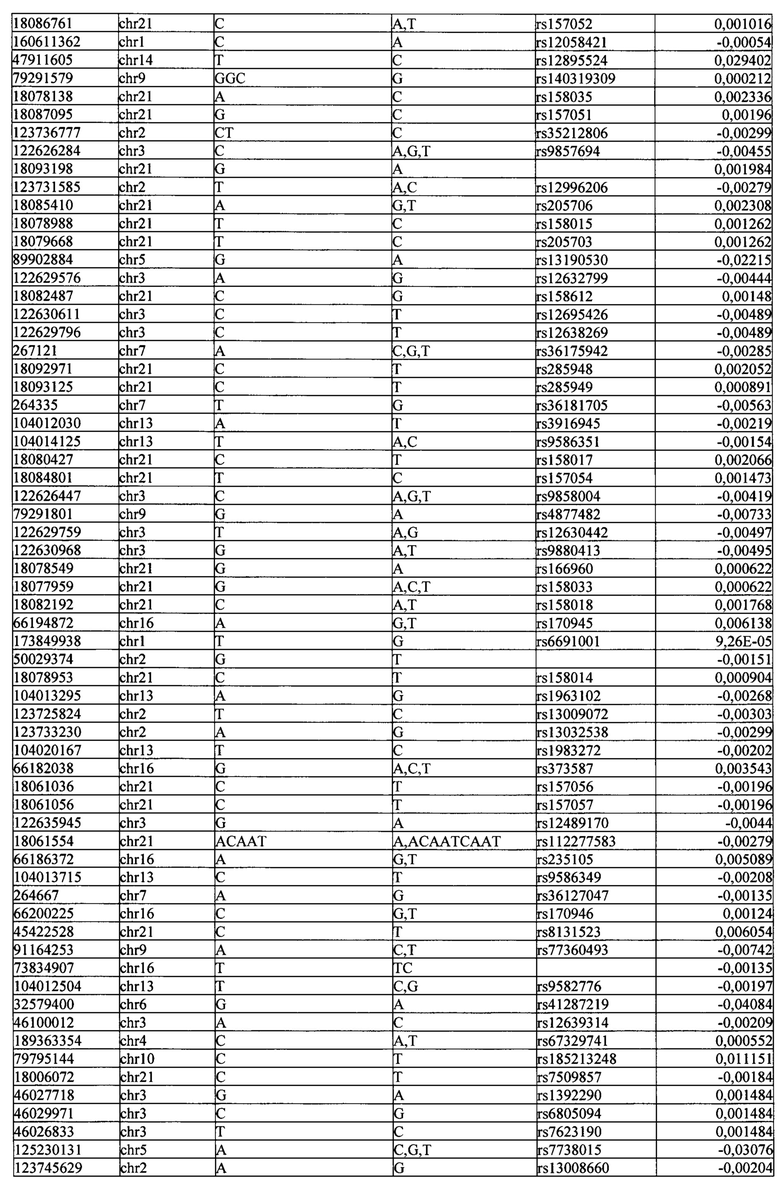
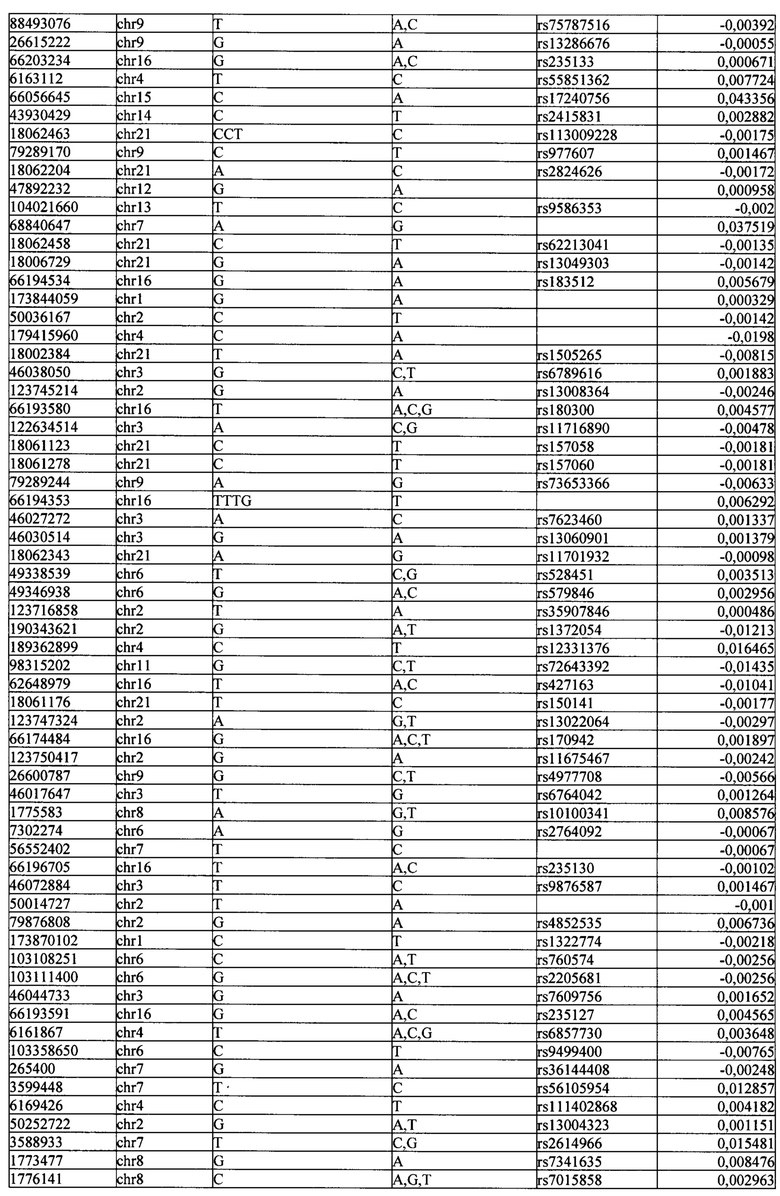
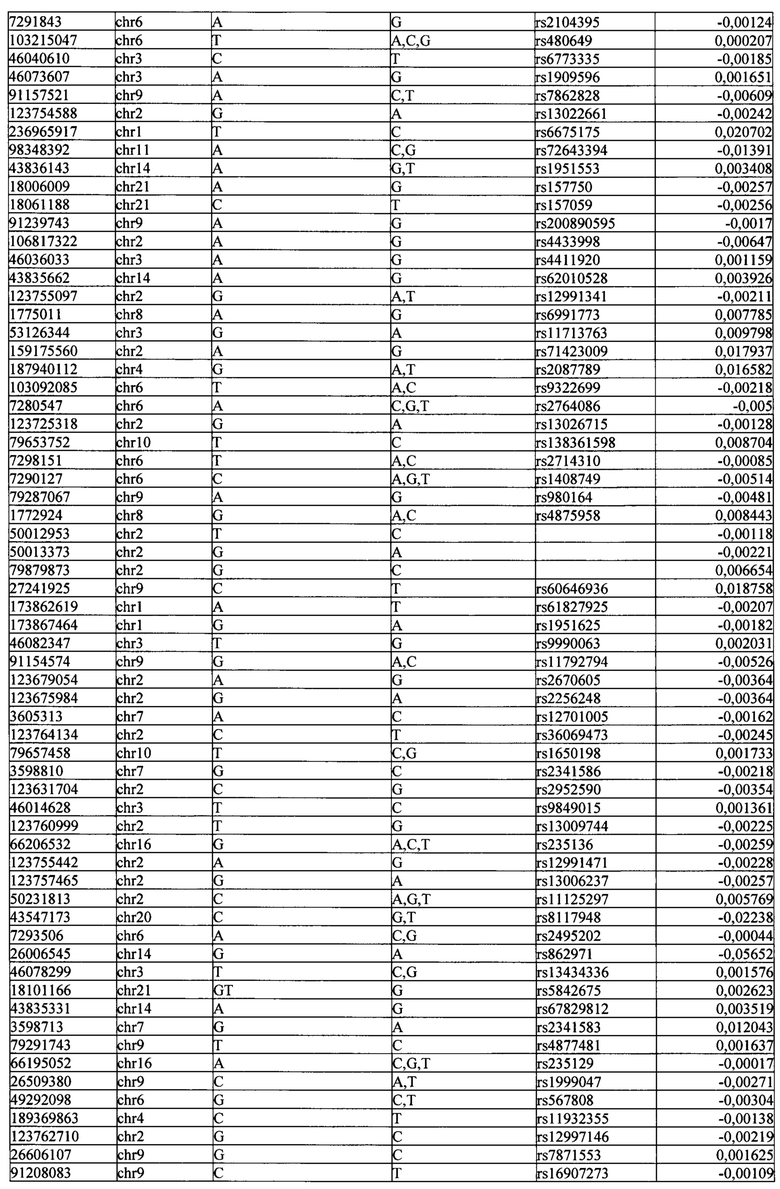
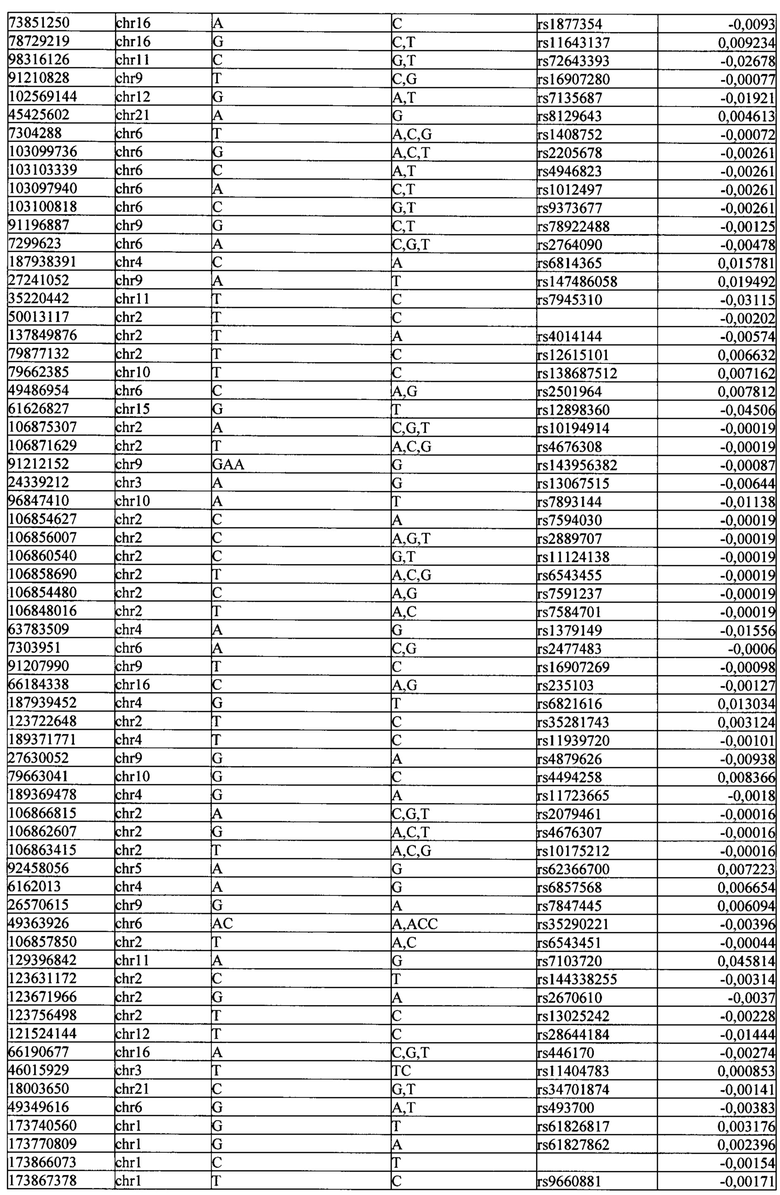
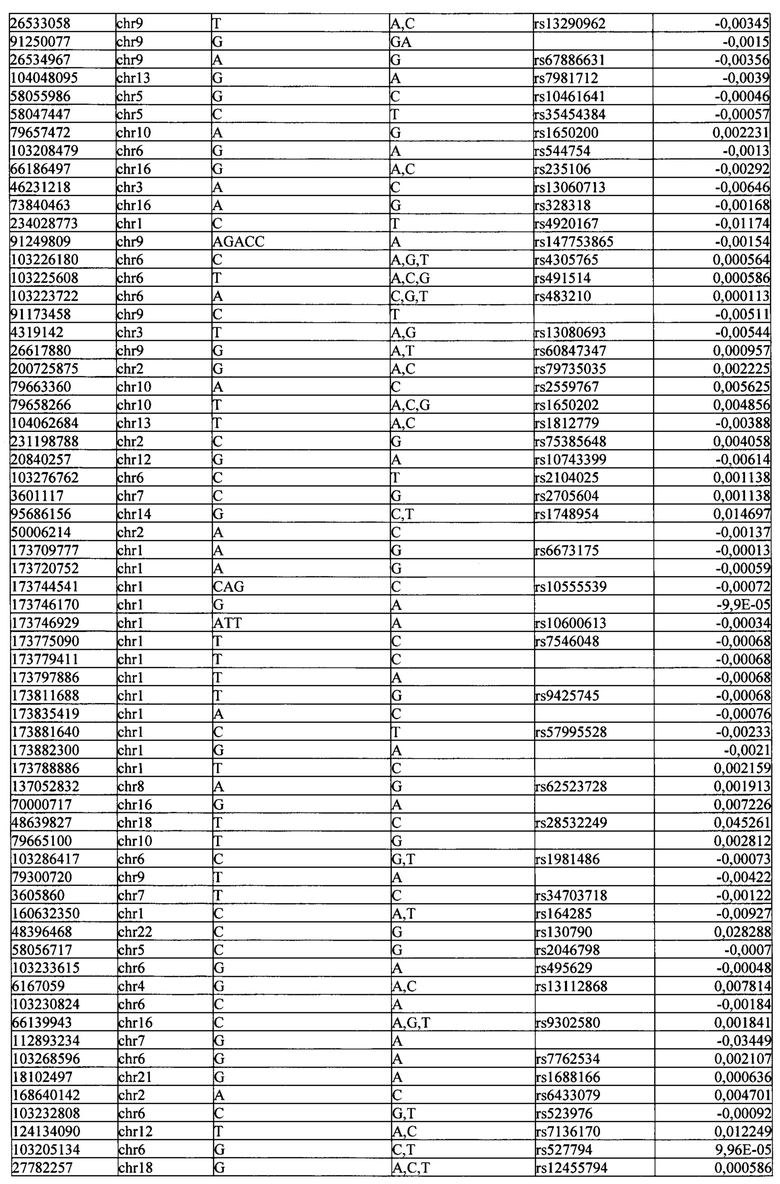
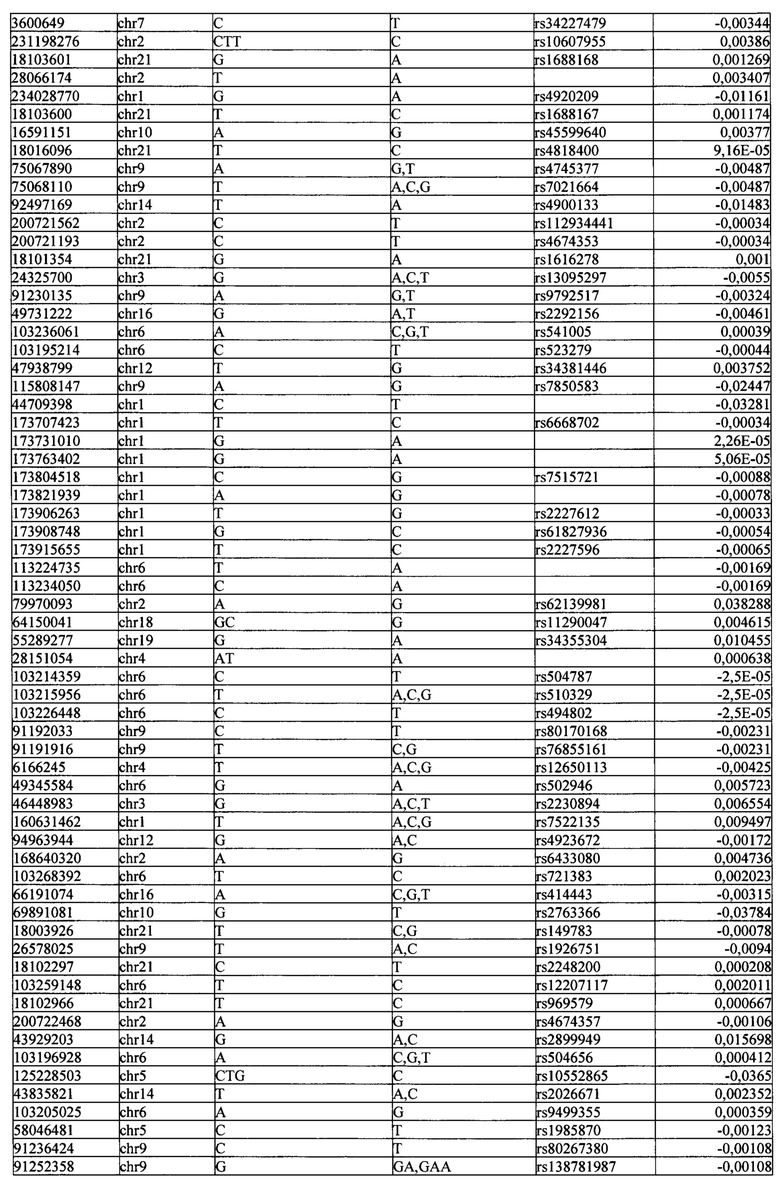
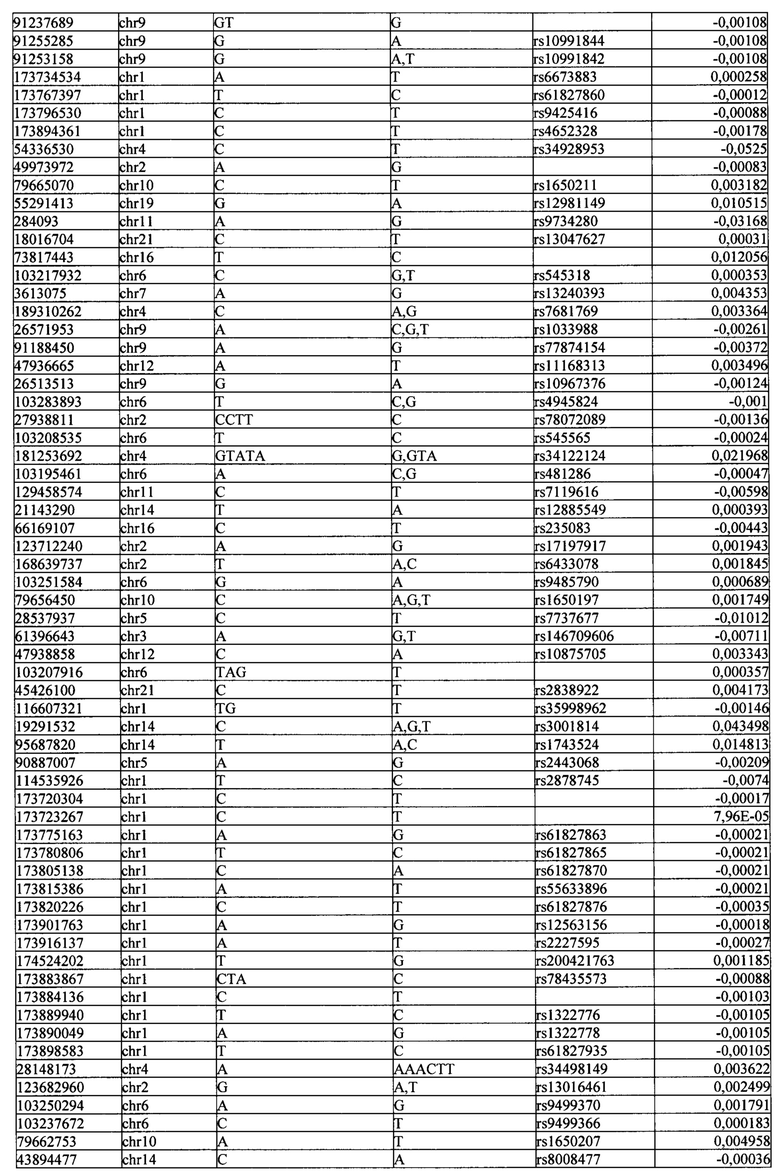
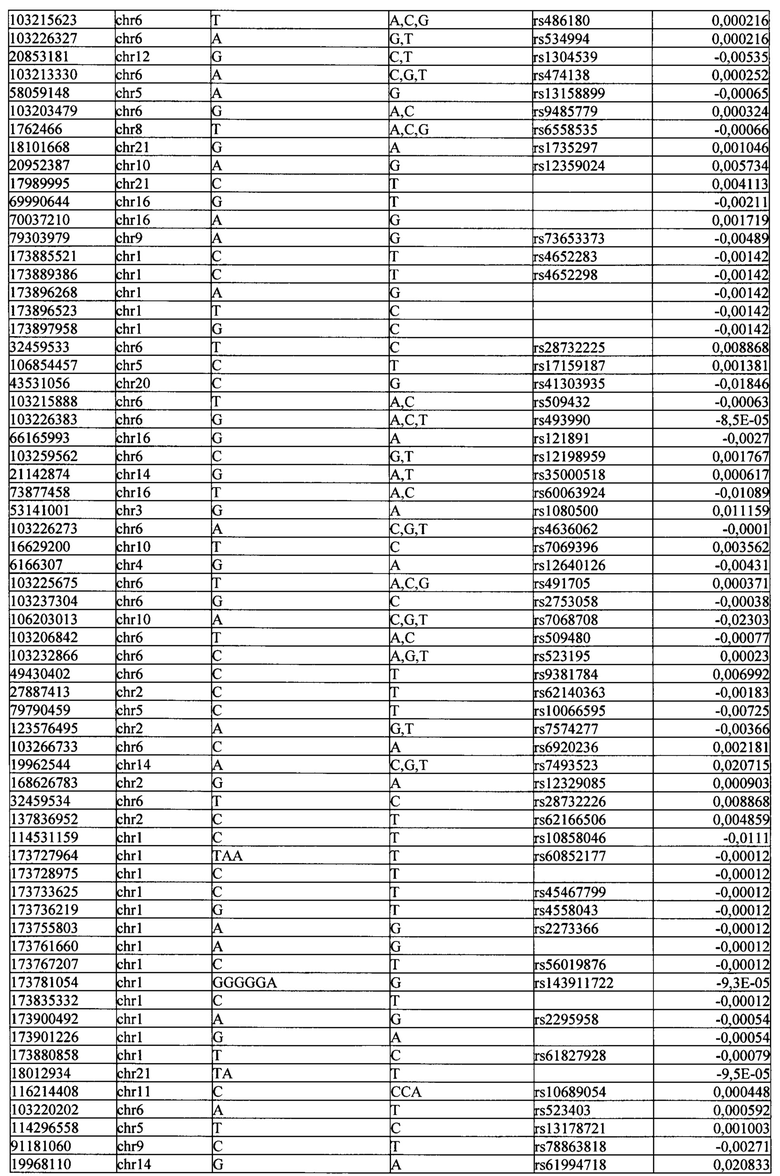
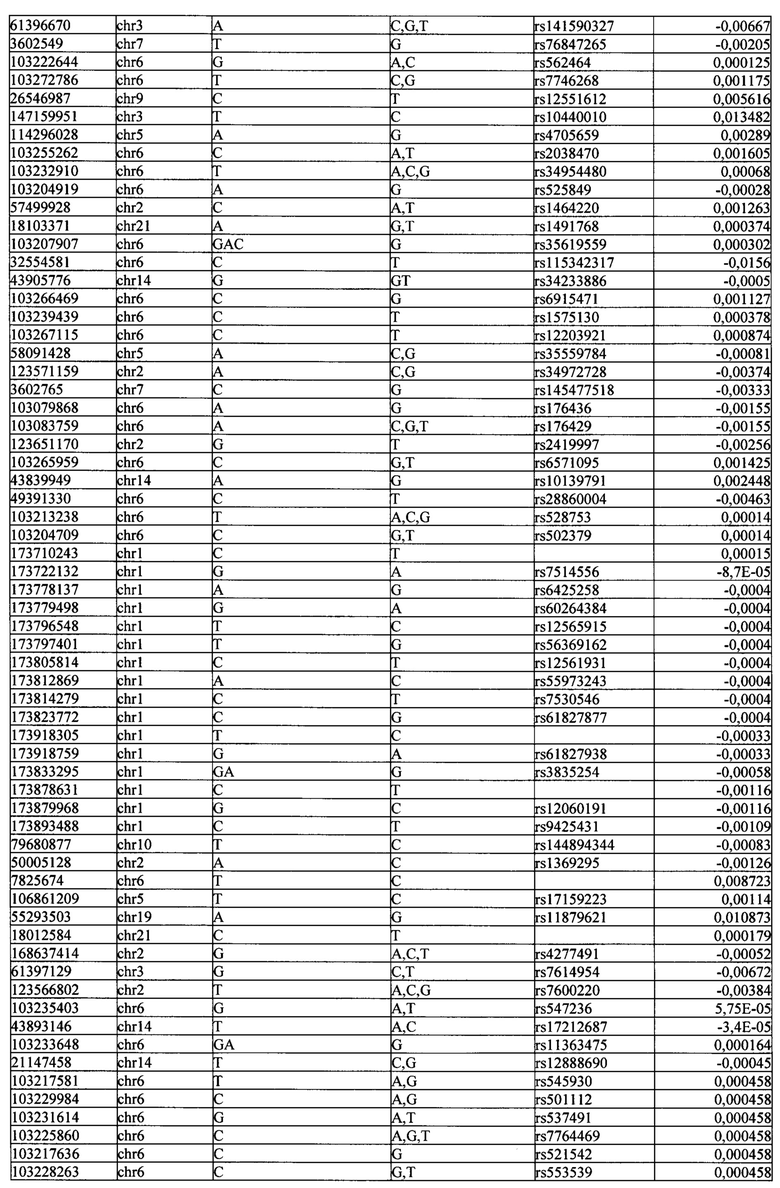
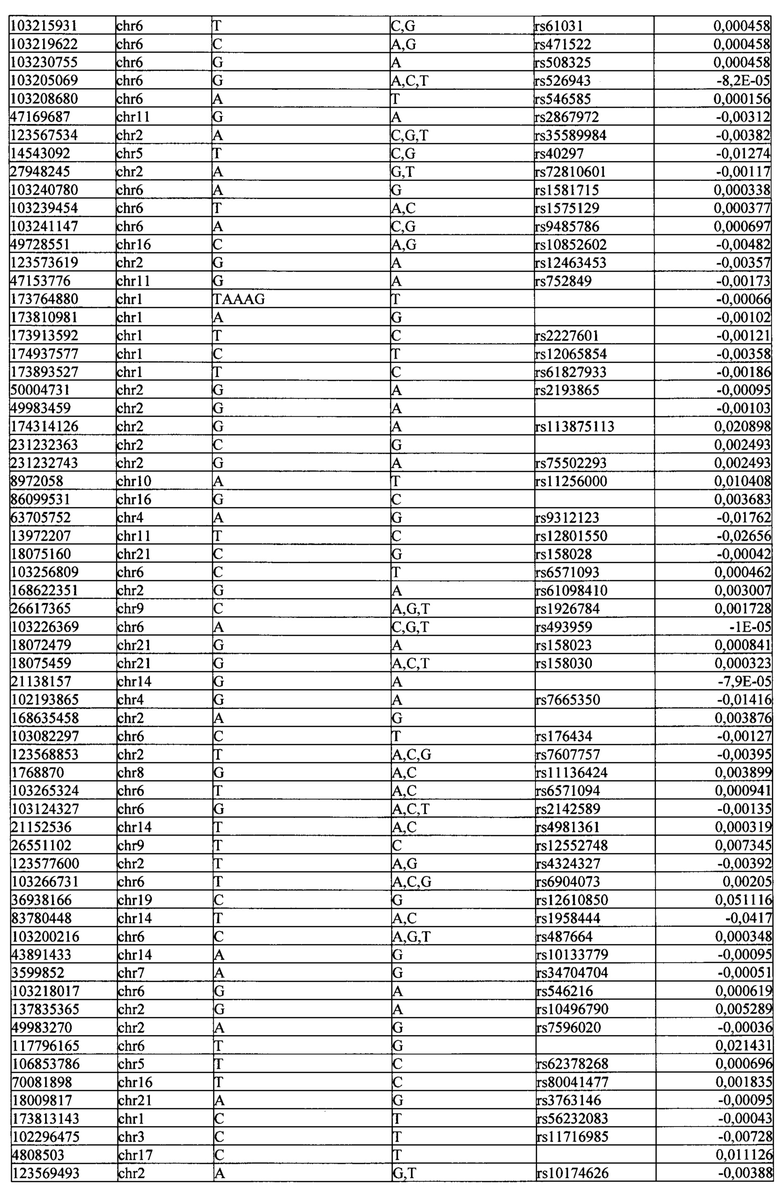
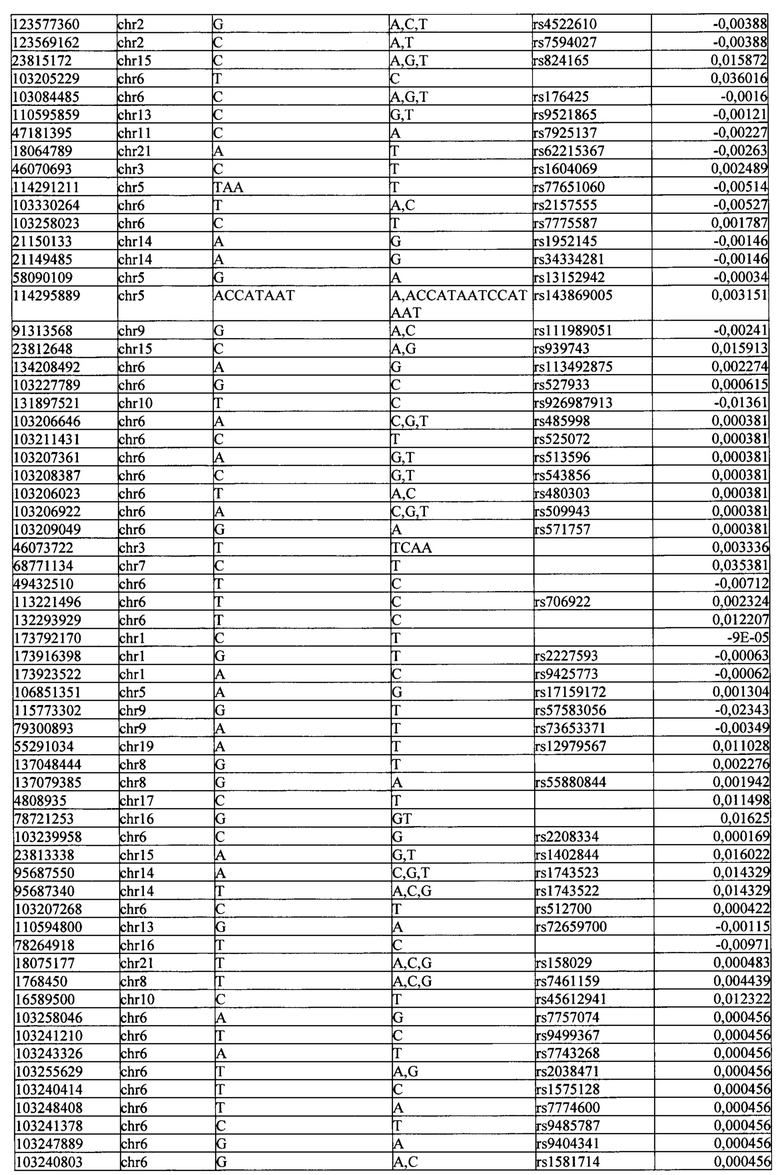
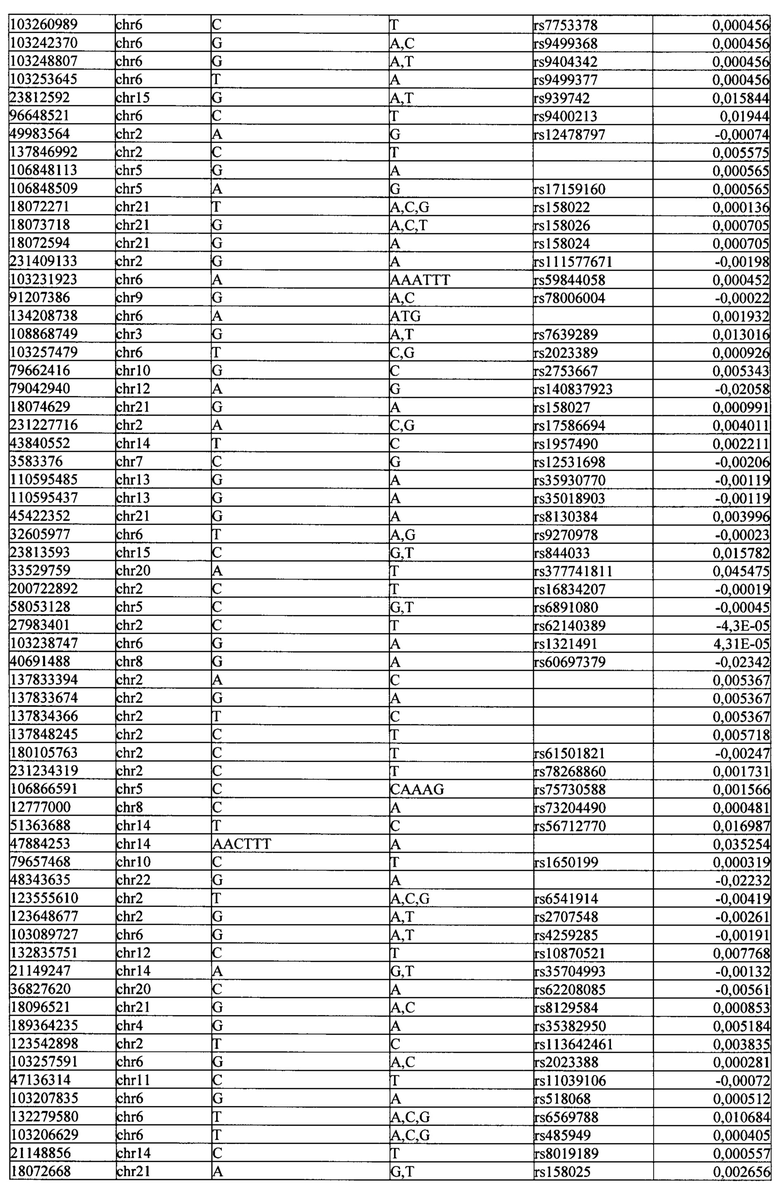
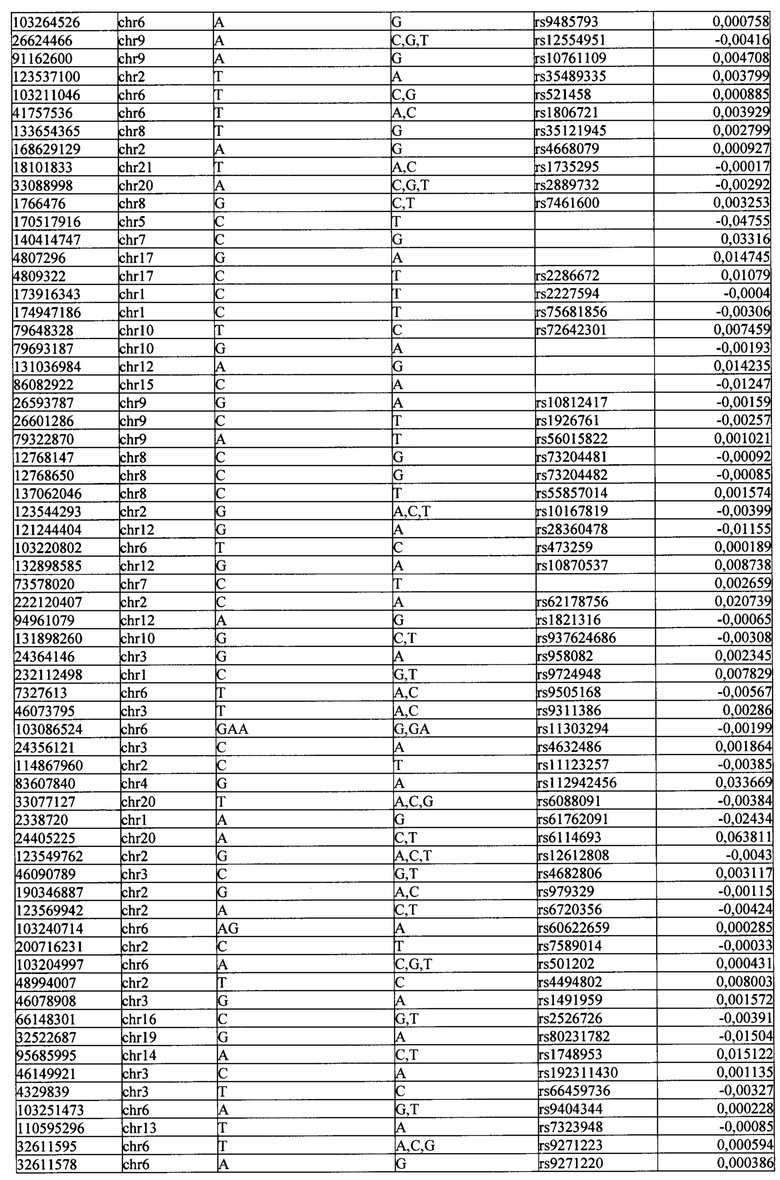
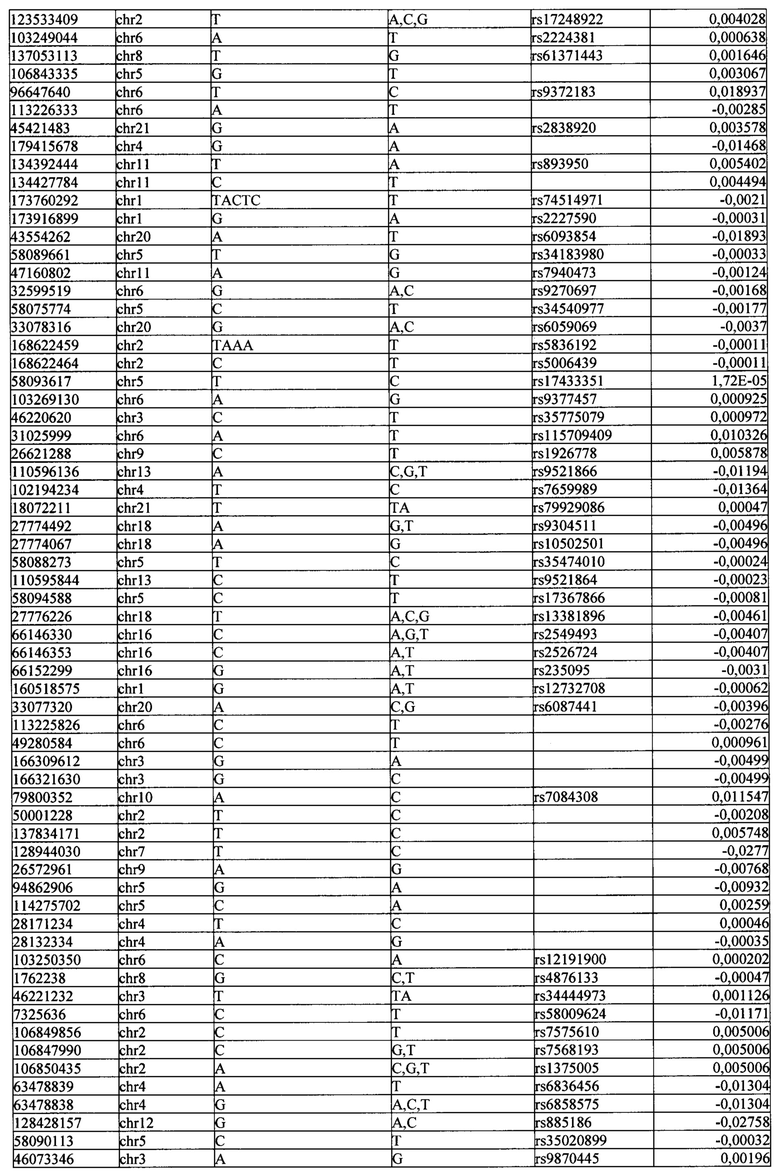
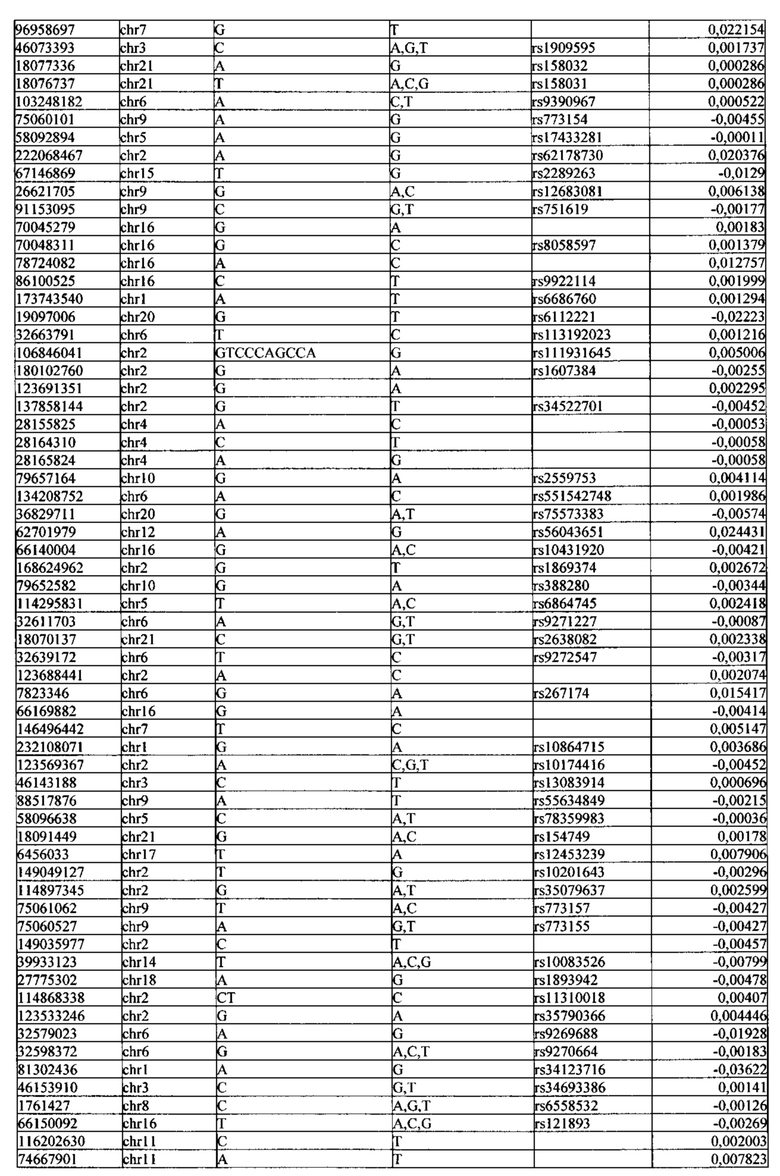
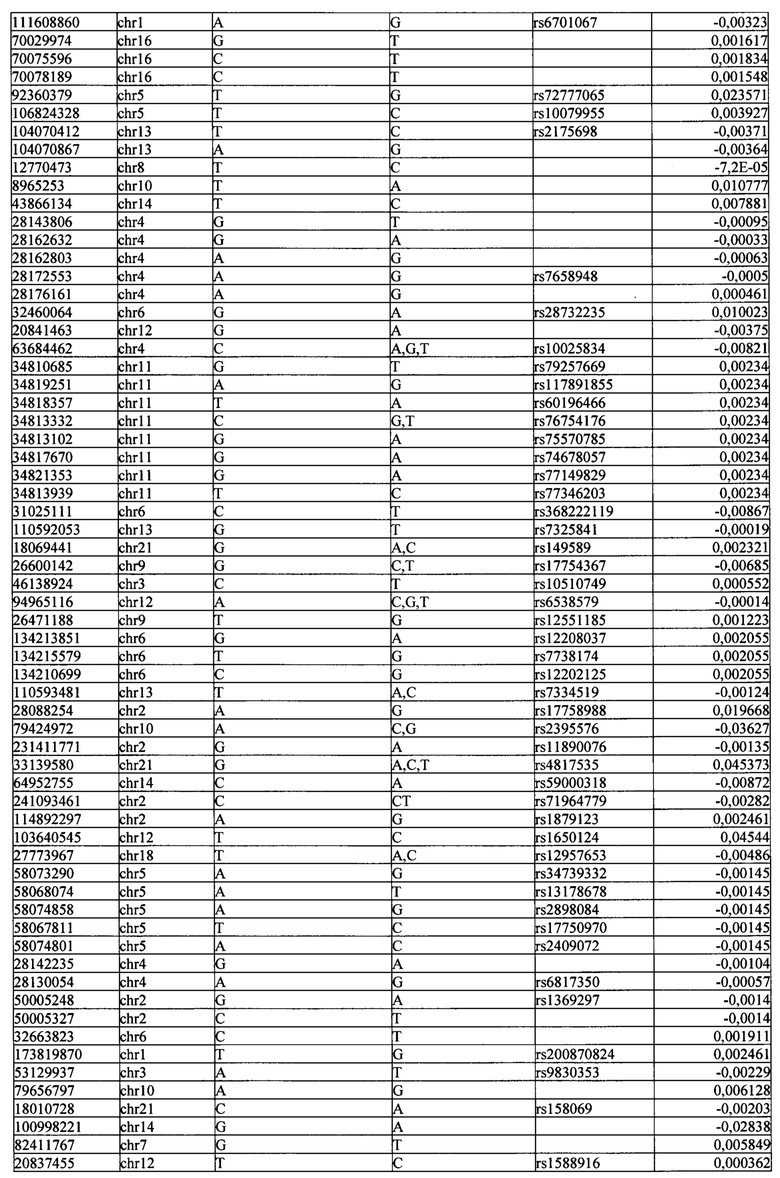
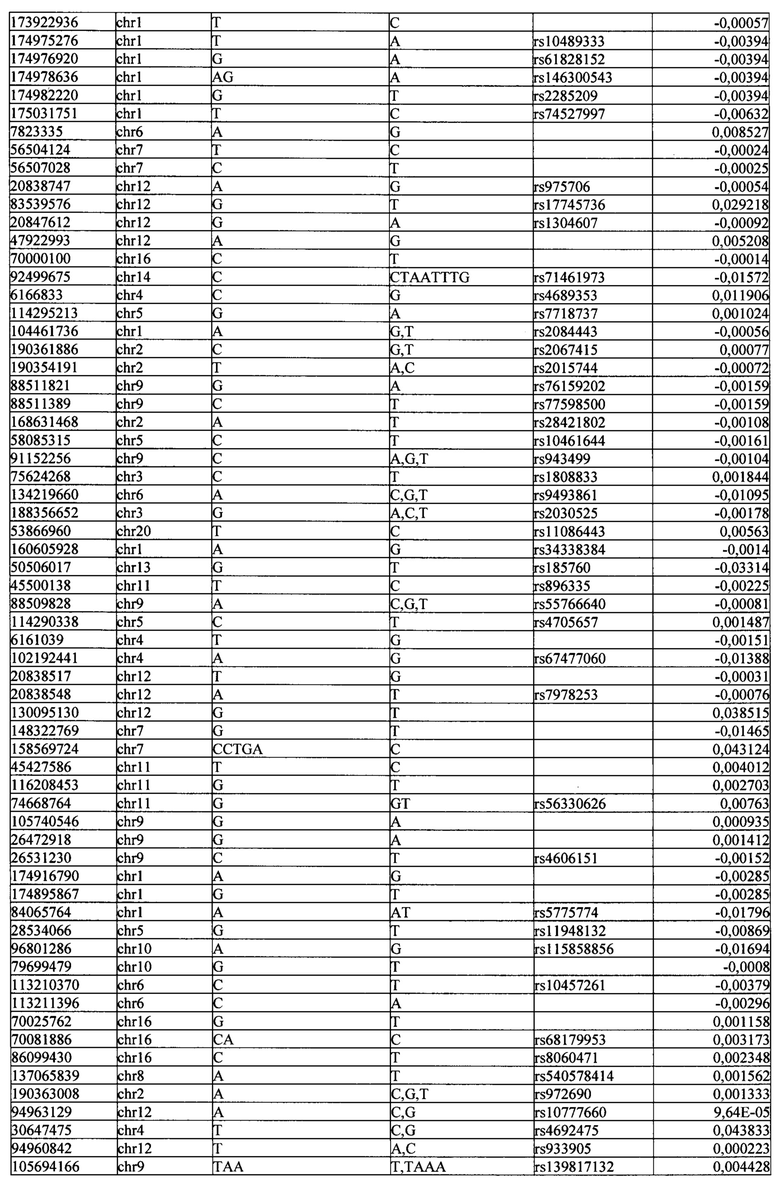
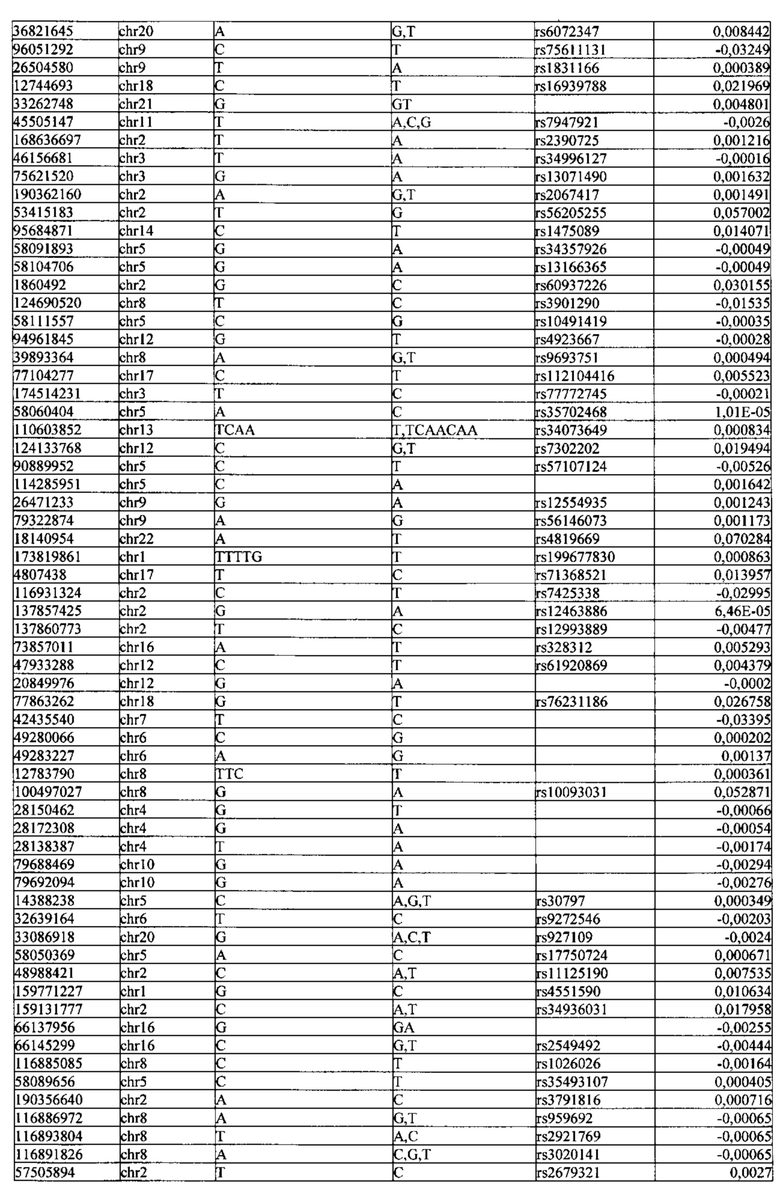
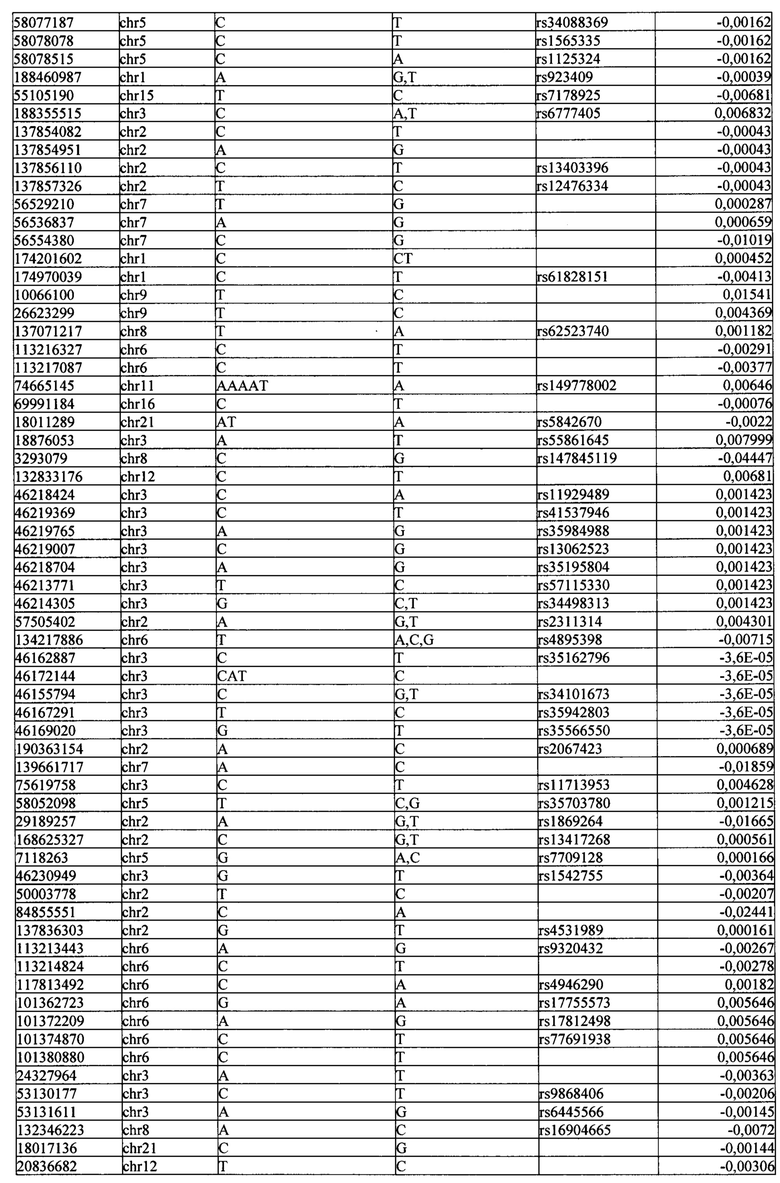
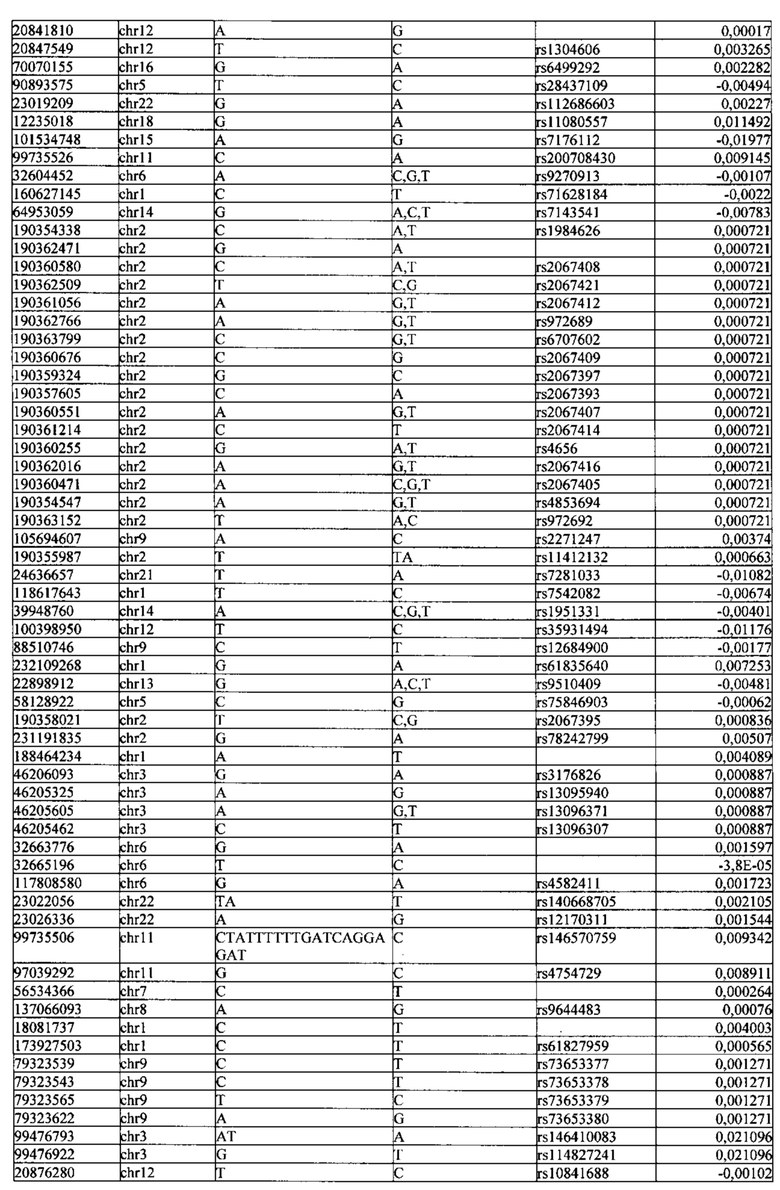
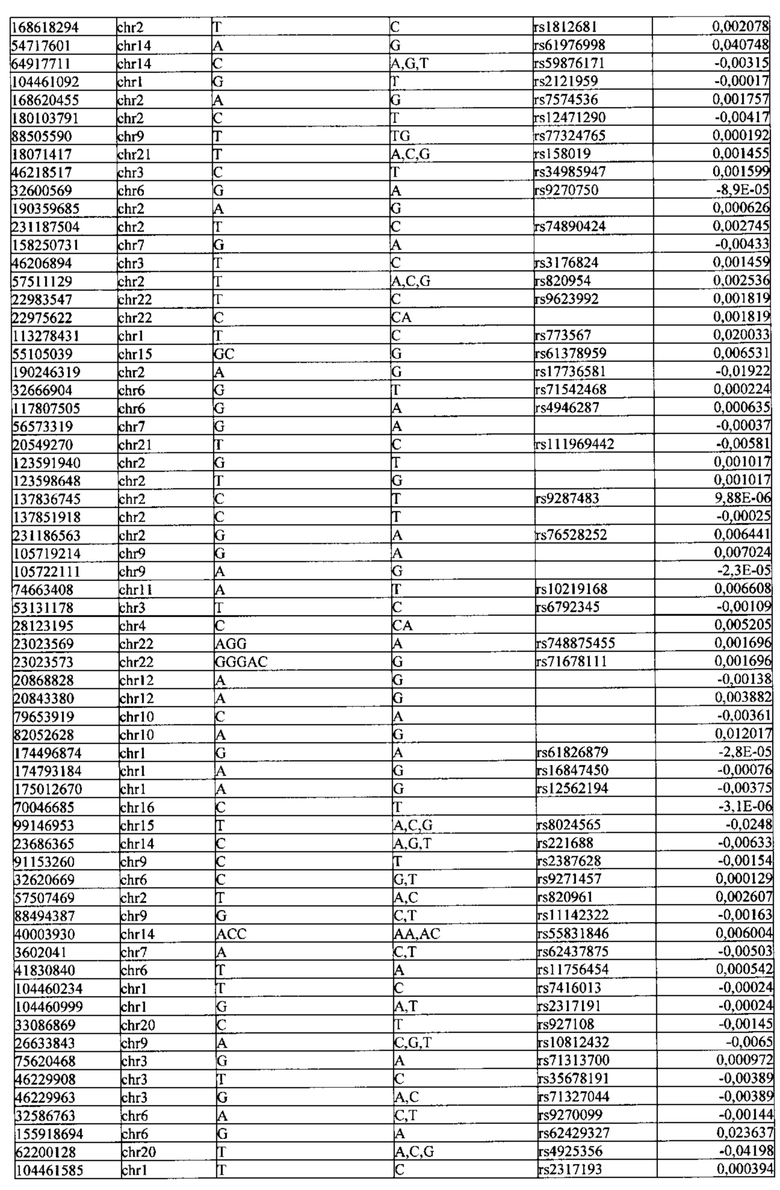
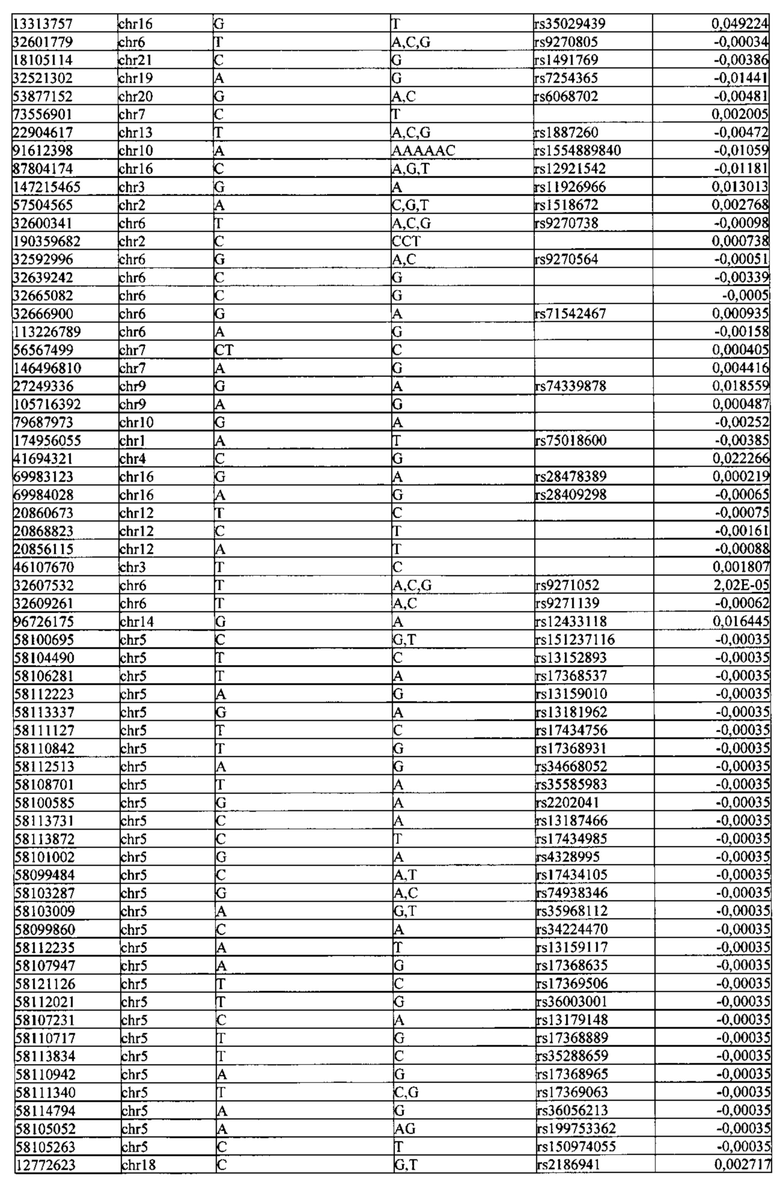
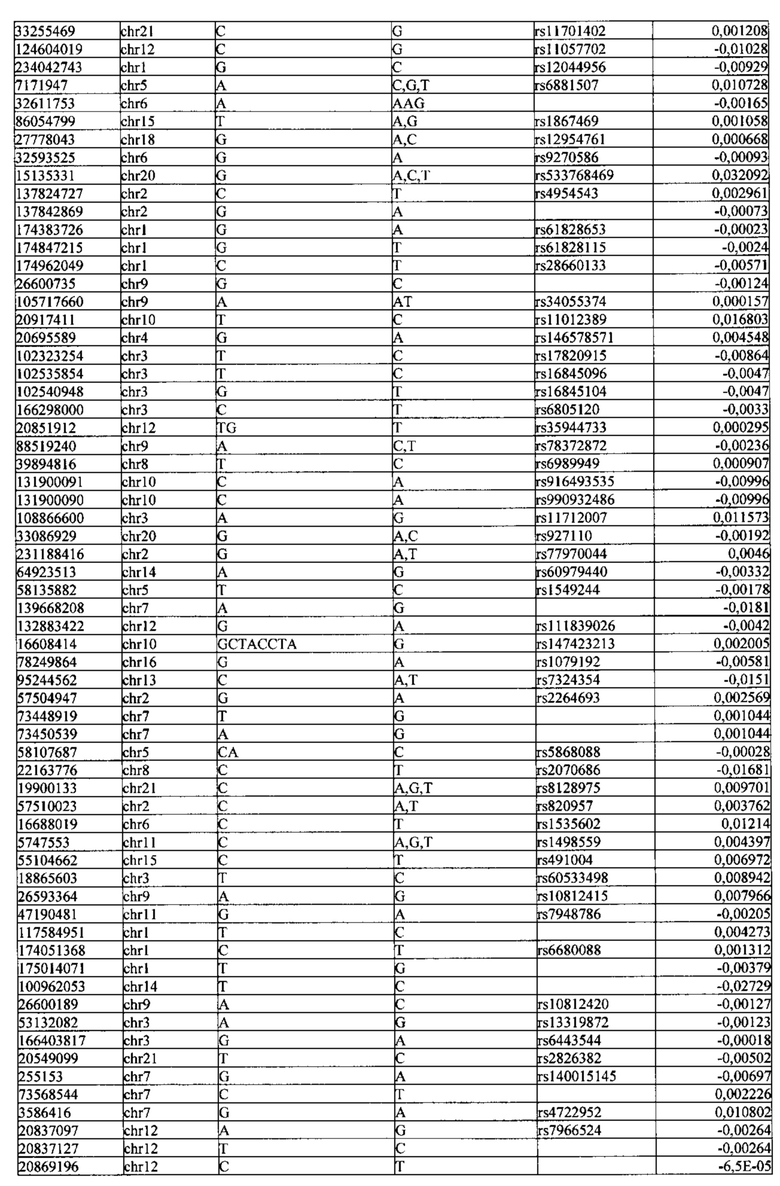
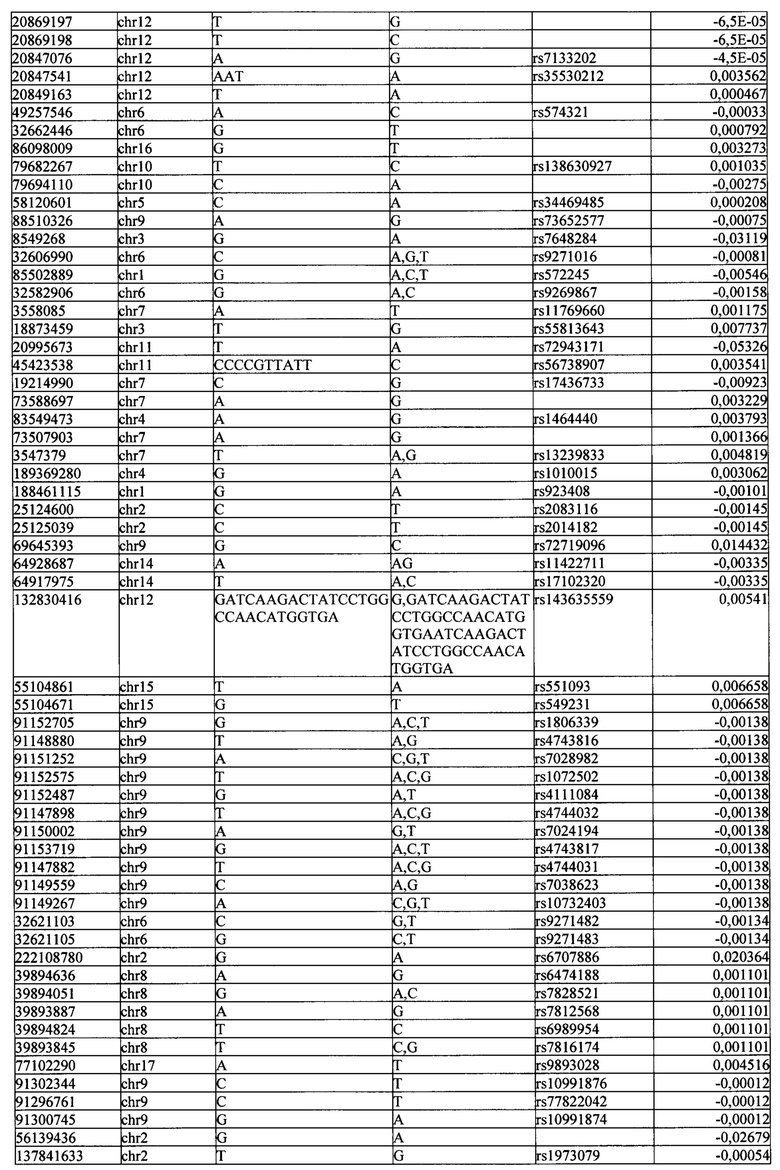
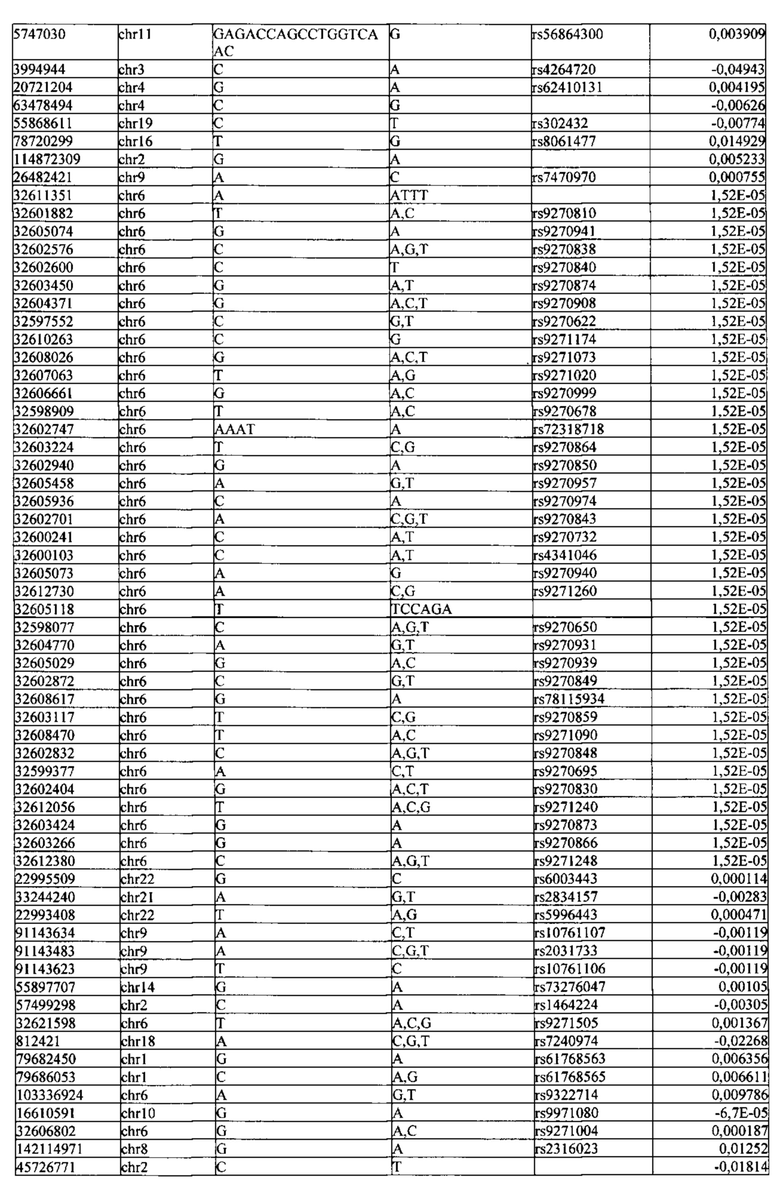
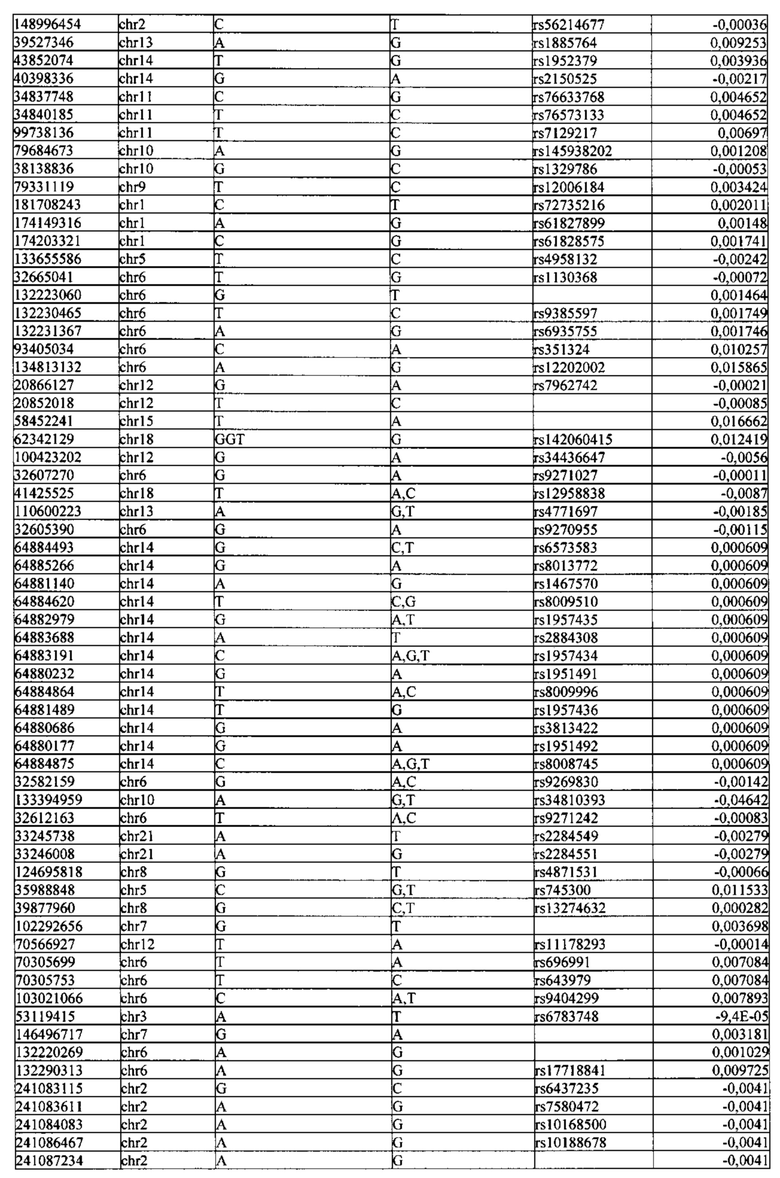
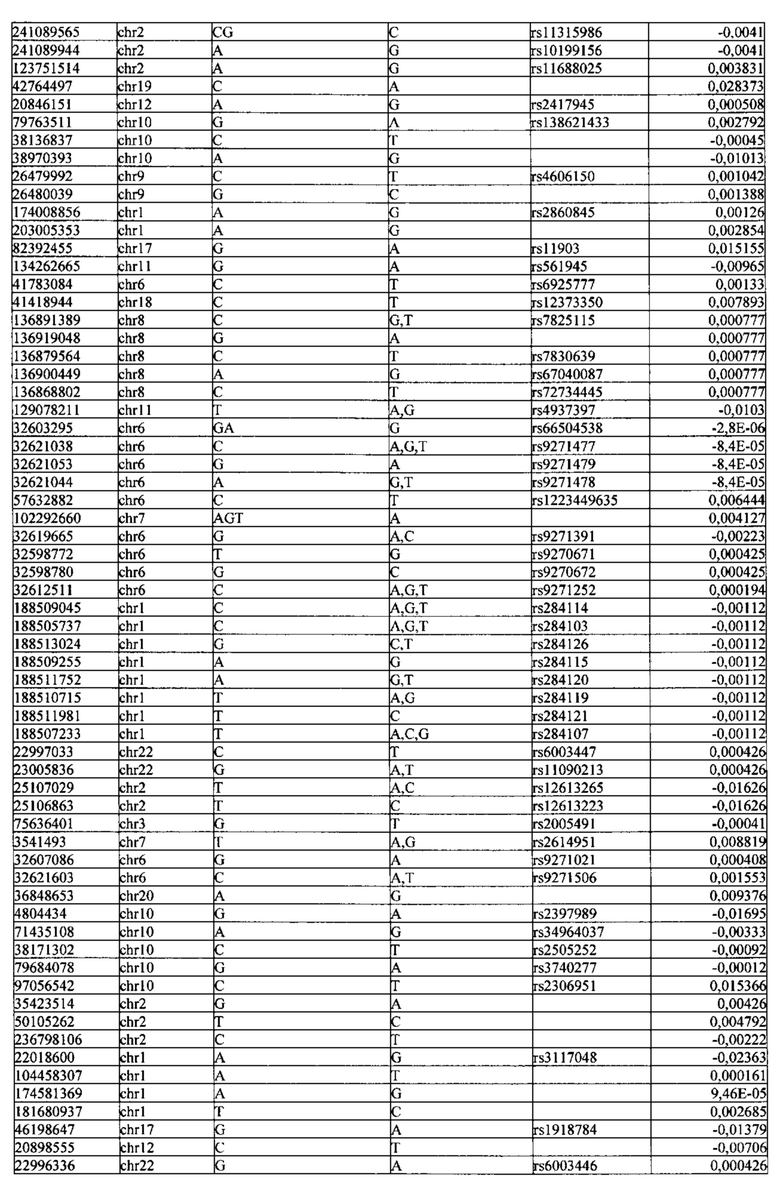
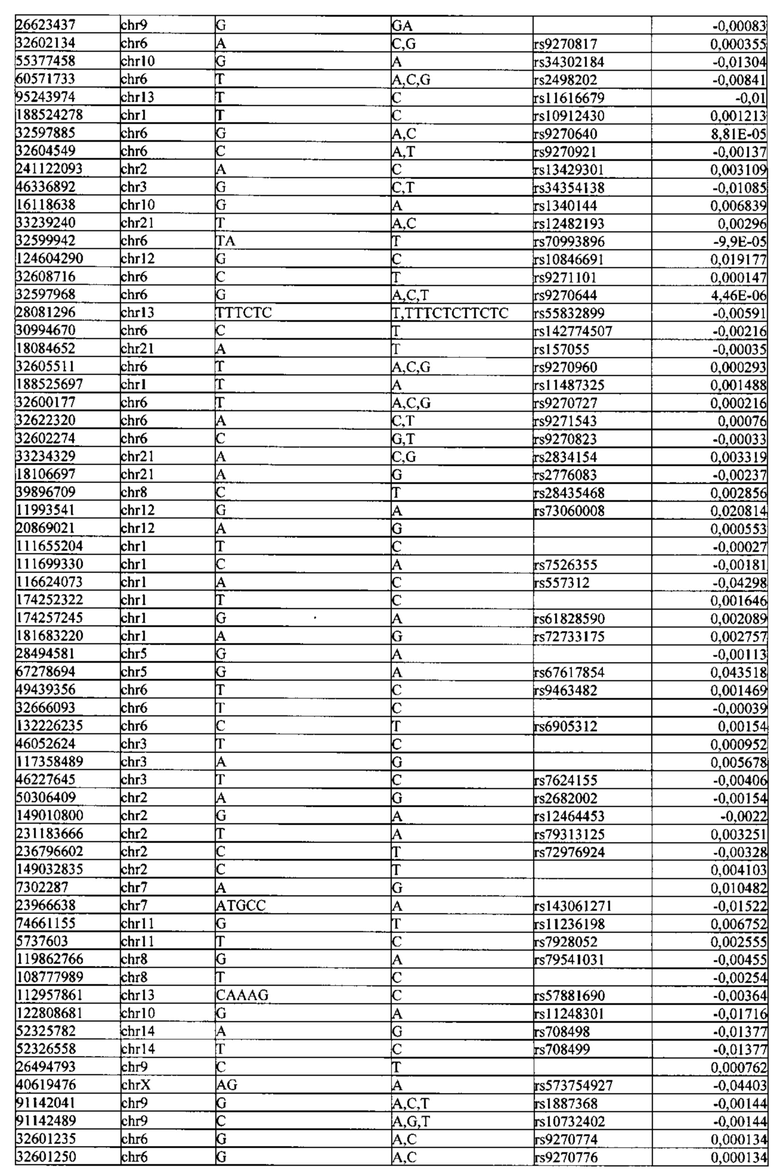

| название | год | авторы | номер документа |
|---|---|---|---|
| Способ прогнозирования риска когнитивных нарушений у долгожителей | 2023 |
|
RU2822168C1 |
| Способ прогнозирования риска развития COVID-19 у больных гемобластозами | 2022 |
|
RU2783422C1 |
| ПРОГНОЗИРОВАНИЕ ПРОГРЕССИРОВАНИЯ ВОЗРАСТНОЙ МАКУЛОДИСТРОФИИ ДО ПОЗДНЕЙ СТАДИИ С ПОМОЩЬЮ ПОЛИГЕННОГО ПОКАЗАТЕЛЯ | 2011 |
|
RU2593954C2 |
| Способ прогнозирования риска развития остеоартроза коленного сустава у мужчин | 2023 |
|
RU2816024C1 |
| Способ прогнозирования риска развития остеоартроза коленного сустава с учетом генетических маркеров | 2023 |
|
RU2804693C1 |
| СПОСОБ ОЦЕНКИ РИСКА НЕОБХОДИМОСТИ ПРИМЕНЕНИЯ КИСЛОРОДНОЙ ТЕРАПИИ ПРИ COVID-19 | 2021 |
|
RU2777487C1 |
| Способ прогнозирования исходов COVID-19 | 2023 |
|
RU2830374C1 |
| СПОСОБ ОЦЕНКИ РИСКА РАЗВИТИЯ ТЯЖЕЛОЙ ФОРМЫ COVID-19 | 2021 |
|
RU2747097C1 |
| СПОСОБ ОЦЕНКИ РИСКА РАЗВИТИЯ ТЯЖЕЛОЙ ФОРМЫ COVID-19 | 2021 |
|
RU2751410C1 |
| СПОСОБ ДИАГНОСТИКИ ТРОМБИНЕМИИ У ПАЦИЕНТОВ С ИНФЕКЦИЕЙ COVID-19 | 2022 |
|
RU2794035C1 |
Изобретение относится к медицине, а именно к инфекционным болезням, и может быть использовано для прогнозирования риска тяжелого течения COVID-19 у пациента. Осуществляют забор крови для полногеномного секвенирования. Выявляют аллели риска тяжелого течения COVID-19. Суммируют коэффициенты однонуклеотидных полиморфизмов (ОНП) из таблицы 2 с выявленными у пациента аллелями риска тяжелого течения COVID-19 по формуле:
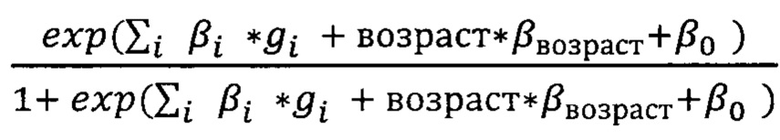 ,
,
где gi - количество аллелей риска тяжелого течения COVID-19, βi - коэффициент для ОНП из таблицы 2, βвозраст - коэффициент регрессии для возрастной переменной, равный 0,02, β0 - коэффициент 0,48 для женщин или 0,71 для мужчин, ехр - экспоненциальная функция. При полученном значении меньше 0,5 прогнозируют легкое течение заболевания. При значении больше 0,5 прогнозируют тяжелое течение заболевания. Способ обеспечивает эффективное прогнозирование риска развития тяжелого течения COVID-19 за счет использования оригинальной расчетной формулы. 2 ил., 3 табл., 2 пр.
Способ прогнозирования риска тяжелого течения COVID-19 у пациента, включающий взятие крови у пациента для полногеномного секвенирования, выявление аллелей риска тяжелого течения COVID-19, суммирование коэффициентов однонуклеотидных полиморфизмов (ОНП) из таблицы 2 с выявленными у пациента аллелями риска тяжелого течения COVID-19 по формуле: 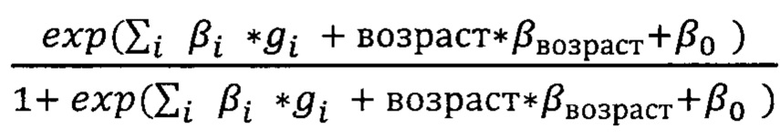 , где gi - количество аллелей риска тяжелого течения COVID-19, βi - коэффициент для ОНП из таблицы 2, βвозраст - коэффициент регрессии для возрастной переменной, равный 0,02, β0 - коэффициент 0,48 для женщин или 0,71 для мужчин, ехр - экспоненциальная функция;
, где gi - количество аллелей риска тяжелого течения COVID-19, βi - коэффициент для ОНП из таблицы 2, βвозраст - коэффициент регрессии для возрастной переменной, равный 0,02, β0 - коэффициент 0,48 для женщин или 0,71 для мужчин, ехр - экспоненциальная функция;
и при значении меньше 0,5 прогнозируют легкое течение заболевания, а при значении больше 0,5 прогнозируют тяжелое течение заболевания.
| СПОСОБ ОЦЕНКИ РИСКА РАЗВИТИЯ ТЯЖЕЛОЙ ФОРМЫ COVID-19 | 2021 |
|
RU2747097C1 |
| СПОСОБ ОЦЕНКИ РИСКА РАЗВИТИЯ ТЯЖЕЛОЙ ФОРМЫ COVID-19 | 2021 |
|
RU2751410C1 |
| WO 2021237292 A1, 02.12.2021 | |||
| PRAKRITHI P | |||
| et al | |||
| Способ изготовления электрических сопротивлений посредством осаждения слоя проводника на поверхности изолятора | 1921 |
|
SU19A1 |
| Front | |||
| Genet | |||
| Способ регенерирования сульфо-кислот, употребленных при гидролизе жиров | 1924 |
|
SU2021A1 |
Авторы
Даты
2023-03-09—Публикация
2022-07-18—Подача