Область техники
Изобретение относится к рекомбинантной плазмиде pET-GST-3CL-GPG, обеспечивающей синтез протеазы 3CL SARS-CoV-2 в клетках E. coli в растворимой форме и может быть использовано в биотехнологии для получения фермента 3CL (с англ.: 3C-like protease), являющегося основной протеазой сарбековируса SARS-CoV-2. Получаемый белок 3CL может быть использован для in vitro поиска ингибиторов основной протеазы SARS-CoV-2 потенциальных терапевтических препаратов (Froggatt, Н.М., Heaton, В.Е., Heaton, N.S. (2020). Development of a fluorescence-based, high-throughput SARS-CoV-2 3CLрrо reporter assay. Journal of virology, 94 (22), e01265-20.; Zhu, W., Xu, M., Chen, C.Z., Guo, H., Shen, M., Нu, X., … & Zheng, W. (2020). Identification of SARS-CoV-2 3CL protease inhibitors by a quantitative high-throughput screening. ACS pharmacology & translational science, 3 (5), 1008-1016.). Способ включает дизайн генетических конструкций, метод трансформации клеток E.coli, очистки и оценки экспрессии целевого белка, метод анализ ферментативной активности.
Уровень техники
Геном SARS-CoV-2 кодирует более двадцати белков, среди которых присутствуют две протеазы - папаин-подобная PLpro и 3-С подобная 3CLрrо (3CL), (или Мрrо, основная протеаза). Обе протеазы играют важную роль в репликативном цикле вируса, их функцией является ферментативный гидролиз двух синтезируемых вирусных полипротеинов (1А и 1АВ) на отдельные функциональные белки.
В силу своих небольших размеров, а также высокой гомологии с аналогичными белками коронавирусов, вызывающих атипичные пневмонии SARS и MERS, основная протеаза SARS-CoV-2 является наиболее охарактеризованной мишенью для потенциальных антивирусных препаратов. Основная протеаза расщепляет полипротеин 1АВ в 11 специфических сайтах. Последовательность распознавания сайта в большинстве случаев состоит из участка цепи (L-Q)-(S-A-G), где расщепляется связь между глутамином и серином. Ингибирование активности основной протеазы приводит к остановке репликации вируса. Среди ферментов человека не известно протеаз с такой же специфичностью расщепления, что может говорить о вероятном специфическом действии и возможной низкой токсичности ингибиторов основной протеазы SARS-CoV-2 (Zhang L, Lin D, Sun X, et al. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors. Science (New York, NY). 2020; 368 (6489): 409-412. doi.10.1126/science.abb3405 Dai W, Zhang B, Su H, et al. Structure-based design of antiviral drug candidates targeting the SARS-CoV-2 main protease. Science. April 2020: eabb4489. doi:10.1126/science.abb4489 Jin Z, Du X, Xu Y, et al. Structure of Mpro from COVID-19 virus and discovery of its inhibitors. Nature. April 2020: 1-9. doi: 10.103 8/s41586-020-2223-y).
Наиболее близким аналогом (прототипом) к заявляемому изобретению является создание конструкции экспрессионной плазмиды основной протеазы SARS; трансформирование плазмиды экспрессии основной протеазы SARS с получением рекомбинантного штамма Escherichia coli; получение с помощью системы экспрессии целевого белка путем культивирования рекомбинантного штамма Escherichia coli в жидкой питательной среде (в культуре LB) с последующим получением очищенной стабильной основной протеазы SARS-CoV-1 с помощью специального метода очистки (заявка на патент Китая CN111979214A, МПК C12N 15/70; C12N 9/50, опубл. 31.08.2020 г.).
Однако данное изобретение имеет ряд недостатков. Так используемый экспрессионный вектор рЕТ28а не имеет в своем составе последовательности GST, для более эффективного транспорта целевого белка в периплазматическое пространство клеток Escherichia coli. Описанная в патенте процедура очистки целевого белка основана на введение в последовательность основной протеазой коронавируса SARS-CoV-1 гистидиновой метки для металл-аффинной хроматографии. Данный метод позволяет получить чистый препарат, но структура полученного белка отлична от нативной. Кроме того, синтезируемый белок является основной протеазой коронавируса SARS-CoV-1, который существенно отличается нуклеотидной и аминокислотной последовательностями от основной протеазы коронавируса SARS-CoV-2.
Раскрытие изобретения
Задачей настоящего изобретения являлась разработка экспрессионной системы и структуры химерного белка GST-3CL SARS-CoV-2, обеспечивающих получение химерного варианта растворимого белка 3CL SARS-CoV-2.
Техническим результатом изобретения является получение экспрессионной генетической конструкции, содержащей нуклеотидную последовательность белка (протеазы) 3CL с оптимизированными кодонами и обеспечивающей синтез и транспорт химерного варианта белка 3CL в периплазматическое пространство E.coli с получением конечного продукта в виде очищенного белка 3CL в растворимой форме.
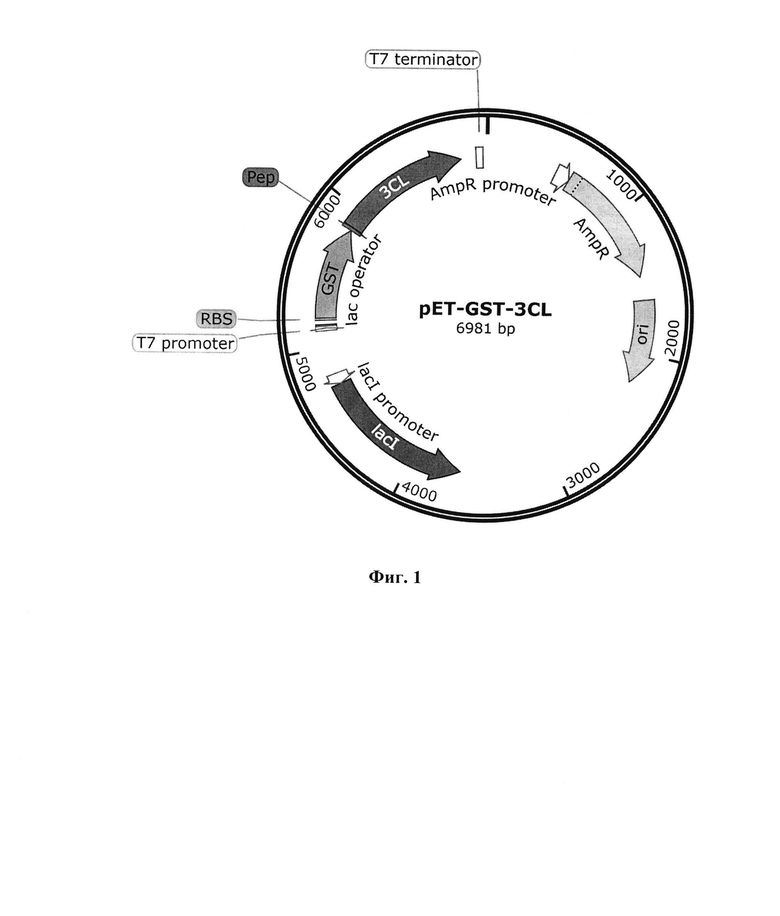
Указанный технический результат достигается тем, достигается тем, что создана рекомбинантная плазмида pET-GST-3CL, обеспечивающая синтез и транспорт химерного белка (протеазы) 3CL SARS-CoV-2 в периплазматическое пространство клеток E. coli в растворимой форме, имеющая нуклеотидную последовательность SEQ ID NO: 2 размером 6981 п.н., и содержащая в соответствии с физической и генетической картой, представленной на фиг. 1, следующие элементы:
- AmpR promoter (координаты с 494 п.н. по 598 п.н.);
- генетический маркер, определяющий устойчивость к ампициллину клеток бактерии E. coli, трансформированных рекомбинантной плазмидой AmpR (координаты с 599 п.н. по 1459 п.н.);
- участок начала репликации ori (координаты с 1630 по 2218 п.н.);
- lacI - ДНК-связывающий белок, который ингибирует экспрессию генов E. coli и обеспечивает экспрессию целевого рекомбинантного белка 3CL после добавления индуктора изопропил-β-D-1-тиогалактопиранозида (IPTG) (координаты с 3648 по 4730 п.н.);
- lacI promoter (координаты с 4731 по 4808 п.н.);
- промотор фага Т7 (координаты с 5117 по 5135 п.н.), обеспечивающий экспрессию последовательности гена GST-3CL в клетках E. coli;
- lac operator (координаты с 5136 по 5160 п.н.);
- RBS - сайт связывания рибосомы (координаты с 5191 по 5196 п.н.);
- ген GST - глутатион-S-трансфераза из Schistosoma japonicum (координаты с 5205 по 5857 п.н.);
- Pep - участок, кодирующий пептид GSTSAVLQ (координаты с 5859 по 5882 п.н.);
- химерный ген 3CL SARS-CoV-2, имеющий нуклеотидную последовательность SEQ ID NO: 1, координаты с 5883 п.н. по 6806 п.н. и кодирующий синтез протеазы 3CL с аминокислотной последовательностью SEQ ID NO: 3;
- Т7 terminator (координаты с 6909 п.н. по 6956 п.н.).
Таким образом, получена кодон-оптимизированная нуклеотидная последовательность, имеющая последовательность SEQ ID NO: 1, кодирующая белок 3CL SARS-CoV-2, а также получена генетическая плазмидная конструкция pET-GST-3CL, представленная на фиг. 1.
Кроме того, получен химерный белок GST-3CL. Последовательность GST на N-конце белка обеспечивает транспорт слитого белка в периплазматическое пространство E. coli. В периплазматическом пространстве происходит автоотщепление GST благодаря каталитической активности 3CL. Это так же достигается включением последовательности GSTSAVLQ между белком GST и 3CL. Последовательность GSTSAVLQ является консенсусной последовательностью сайта протеолиза 3CL.
Белок 3CL с аминокислотной последовательностью SEQ ID NO: 3, получают культивированием клеток E. coli, синтезом целевого белка и выделением растворимого белка с помощью ионообменной хроматографии.
Изобретение имеет ряд преимуществ по сравнению с близкими по сущности аналогами. Использование белка GST позволяет получить белок 3CL в растворимой и нативной форме, сохраняющей ферментативную активность, что позволяет использовать его для анализа веществ ингибиторов коронавируса SARS-CoV-2. Проведенная оптимизация кодонов фрагмента ДНК, кодирующего белок 3CL, обеспечивает высокий уровень экспрессии белка. Последовательность GSTSAVLQ обеспечивает эффективное автоотщепление белка 3CL. Отсутствие в составе белка дополнительных аминокислотных последовательностей позволяет сохранить нативные свойства вирусной протеазы SARS-CoV-2.
Осуществление изобретения
Изобретение иллюстрируется следующими графическими материалами.
На фиг. 1. Представлена физическая карта плазмидного вектора рЕТ-GST-3CL, имеющего размер 6981 п.н.
На фиг. 2 приведена электрофореграмма разделения препаратов белков в 15% ДСН-ПААГ:
1 - биомасса E. coli до добавления индуктора IPTG;
2 - биомасса E. coli после добавления индуктора IPTG;
3 - фракция белков периплазмы (растворимая);
4 - нерастворимая фракция белков;
М - маркер молекулярного веса, 10-200 кДа
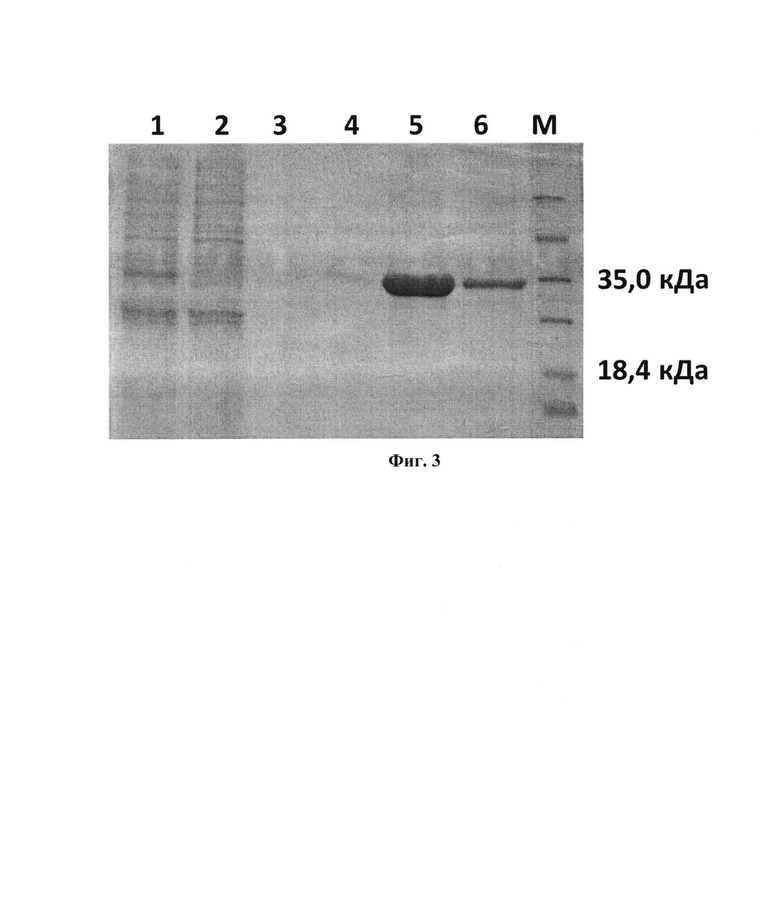
На фиг. 3 приведена электрофореграмма разделения хроматографических фракций в 15% ДСН-ПААГ:
1 - лизат клеток E. coli содержащих плазмиду pET-GST-3CL до нанесения на колонку;
2-6 - фракции белка при элюировали в линейном градиенте концентрации NaCl от 0 до 1 М в 20 мМ Трис-НСl;
М - маркер молекулярного веса, 10-200 кДа
Ниже приведены примеры 1-3 конкретного осуществления изобретения.
Пример 1. Конструирование генетической последовательности для синтеза белка 3CL
Последовательность гена, кодирующего белок 3CL (3264S - 3569Q) коронавируса SARS-CoV-2 (GenBank MZ149976) заимствовали из базы данных GenBank и проводили оптимизацию частот встречаемости кодонов для млекопитающих при помощи сервиса JCat (http://www.jcat.de/), при этом индекс адаптации кодонов CAI достиг 0,87.
На основе вектора pET-GST получали конструкцию pET-GST-3CL (фиг. 1), содержащую нуклеотидную последовательность 3CL (SEQ ID NO: 1).
Нуклеотидную последовательность кодирующую пептид Pep (GSTSAVLQ), и сайты рестрикции ВаmHI и CciNI вводили в последовательность 3CL одновременно с помощью ПНР. Использовались пары праймеров 3CL-F и 3CL-R (таблица 1).

Пример 2. Создание плазмидной конструкции (pET-GST-3CL). 2.1 Амплификация методом ПЦР нуклеотидной последовательности, кодирующей 3CL SARS-CoV-2
Амплификацию фрагментов проводили по стандартному протоколу. Реакционная смесь, объемом 50 мкл содержала 10×буфер, 25 мМ dNTP, олигонуклеотидные праймеры (таблица 1) в количестве 15 пмоль каждого, 5 ед. QS-полимеразы, 1 мкл раствора плазмидной ДНК. Реакцию проводили в амплификаторе Veriti™ 96-Well Thermal Cycler. Температурно-временной профиль ПЦР: плавление - 95°С - 5 мин. (1 цикл). 95°С - 30 секунд. Отжиг - 58°С - 30 секунд, элонгация - 72°С - 30 секунд (35 циклов).
2.2. Выделение ДНК из агарозного геля
После визуализации под УФ-излучением фрагменты ДНК необходимой длины, разделенные в агарозном геле, вырезали из геля и элюировали при помощи набора «Евроген» Cleanup Standard (Россия) в соответствии с рекомендациями производителя.
2.3. Ферментативный гидролиз вектора pET-GST и ПЦР-продукта (последовательность Pep-3CL)
Для клонирования гена 3CL в составе вектора pET-GST были использованы эндонуклеазы рестрикции ВаmHI и CciNI (фирма «Сибэнзим», Россия) с прилагаемыми к ним буферами. Реакционную смесь готовили в соответствии с активностью фермента (2-5 е.а. на 1 мкг ДНК) и концентрации плазмидной ДНК. Условия реакции: температура, состав буфера и длительность проведения ферментативного гидролиза ДНК подбирали в соответствии с инструкциями производителя "Сибэнзим".
2.4. Лигирование ПЦР-продукта (последовательность Pep-3CL) и вектора pET-GST
Реакцию лигирования проводили в течение 30 мин. при комнатной температуре, используя смесь из 2 мкг ампликонов с ДНК-матрицы, 1 мкг векторной плазмиды и 20 е.а. ДНК-лигазы фага Т4 в прилагаемом к коммерческому набору реакционном буфере. Полученную лигазную смесь использовали для трансформирования культуры компетентных клеток Е. coli штамм Neb Stable.
2.5 Heat-shock трансформация
К «компетентным» клеткам Е. coli штамм Neb Stable добавляли 10 мкл лигазной смеси (отношение 1:10), инкубировали на льду в течение 30 мин. После этого клетки подвергали «температурному шоку» при 42°С в течение 45 сек. Охлаждали клетки на льду в течение 2 мин, затем добавляли 200 мкл среды SOB и инкубировали при 37°С в течение 60 мин. По окончании инкубации трансформированные клетки высевали на чашку Петри с твердой питательной средой LB, содержащей ампициллин в концентрации 100 мкг/мл.
2.6 Отбор клонов для рестрикционного анализа
Клетки Е. coli штамма Neb Stable, трансформированные плазмидой селективно культивировали в 10 мл жидкой питательной среды LB с добавлением антибиотика в рабочей концентрации 25 мкг/мл.
2.7 Определение нуклеотидной последовательности ДНК
Секвенирование проводили по методу Сэнгера. Использовался набор CEQ2000Dye Terminator Cycle Sequencing Kit и 16-капилярный автоматический секвенатор ABI 3130×1.
Пример 3. Наработка рекомбинантного белка 3CL
Полученной плазмидой pET-GST-3CL трансформировали клетки Е. coli BL21(DE3). Индивидуальные колонии Е. coli, содержащие рекомбинантные плазмиды, культивировали в течение ночи на орбитальном шейкере в среде LB, содержащей 100 мкг/мл ампициллина, при 37°С и 180 об/мин. Инокулят в соотношении 1/100 переносили в колбу Эрленмейера, содержащую среду LB, и растили до поглощения 0,8 (при λ=600 нм). Добавляли индуктор изопропил-β-D-1-тиогалактопиранозид (ИПТГ) до конечной концентрации 1 мМ. Культуру дополнительно культивировали на шейкере в течение 5 ч при 37°С и 180 об./мин. Биомассу осаждали центрифугированием в течении 20 мин при 5000 g и 4°С. Полученный осадок растворяли в лизирующем буфере: 12 мМ Tris; 120 мМ NaCl; 0.1 mM EDTA; ImM DTT. Для разрушения клеточной стенки проводили ультразвуковую обработку полученного раствора. После суспензию центрифугировали при 16000 g 20 минут при 4°С для отделения нерастворимой фракции. Полученные фракции анализировали при помощи электрофореза в денатурирующих условиях в 15% ПААГ. Белок находился в растворимой фракции (фиг. 2).
Пример 4. Очистка рекомбинантого белка
Супернатант, содержащий целевой белок подвергали хроматографической очистке. Очистку проводили последовательной хроматографией с промежуточным диализом в 20 мМ Трис-HCl, рН 7,6 на колонках с катионообменными сорбентами (SP-сефароза и Nuvia HR-S), уравновешенными 20 мМ Трис-HCl, рН 7,6. Целевую фракцию элюировали в линейном градиенте концентрации NaCl от 0 до 1 М в 20 мМ Трис-HCl, рН 7,6 и анализировали при помощи электрофореза в денатурирующих условиях в 15% ПААГ. Конечный препарат диализовали против 20 мМ Трис-HCl, рН 7,6 и подвергали стерилизующей фильтрации через фильтры с размером пор 0,22 мкМ. Полученные фракции анализировали при помощи электрофореза в денатурирующих условиях в 15% ПААГ (фиг. 3).
Пример 5. Анализ ферментативной активности рекомбинантого белка
Анализ ферментативной активности полученного белка провоидили методом флуориметрии с использованием планшетного флуориметра CLARIOstar Plus, длины волн возбуждения - 495 нм, эмиссии - 520 нм. В качестве субстрата использовали синтетический пептид Dabcyl-VNSTLQSGLR-Lys(5FAM)-MA. Гидролиз субстрата 3CL протеазой приводит к разгоранию флуоресценции из-за удаления флуорофора и тушителя. Скорость увеличения флуоресценции отражает скорость ферментативной реакции.
Реакционные смеси готовили в 384 луночном планшете, после чего инкубировали при комнатной температуре в течение 5 мин. Все измерения проводили при 25°С. Каждая лунка содержала одну реакционную смесь. Калибровку прибора проводили по раствору пептида, подвергшегося полному гидролизу. Значение флуоресценции данной смеси принимали за 80%. Реакционные смеси содержали 5 мкл Tris-HCl буфера (рН 8,0; 50 мМ Tris; 50 мМ NaCl), 2 мкл субстрата (С=33 мкг/мл). Реакцию запускали добавлением 3 мкл 3CL (С=0,025 мг/мл). О ферментативной активности свидетельствовало увеличение сигнала флуоресценции.
При инкубировании специфического пептидного субстрата (Dabcyl-VNSTLQSGLR-Lys(5FAM)-MA) с ферментом протеазой 3CL, происходило увеличение уровня флуоресценции до 200000 RFU (относительных единиц флуоресценции), при инкубации пептидного субстрата (Dabcyl-VNSTLQSGLR-Lys(5FAM)-MA) без добавления фермента регистрируемый уровень флуоресценции не изменяется. Это позволяет судить о специфической ферментативной активности белка 3CL.
--->
Перечень последовательностей
<110> Федеральное бюджетное учреждение науки «Государственный научный центр
вирусологии и биотехнологии «Вектор» Федеральной службы по надзору в
сфере защиты прав потребителей и благополучия человека (ФБУН ГНЦ ВБ
«Вектор» Роспотребнадзора)
<120> Рекомбинантная плазмида pET-GST-3CL, обеспечивающая синтез протеазы
3CL SARS-CoV-2 в клетках E.coli в растворимой форме.
<160> SEQ ID NO:3
<210> SEQ ID NO:1
<211> 924
<212> DNA
<213> Artificial Sequence
<220>
<223> Нуклеотидная последовательность гена 3CL
<400> 1
1 AGCGGTTTTC GTAAAATGGC ATTTCCGAGC GGTAAAGTTG AAGGTTGTAT GGTTCAGGTT
61 ACCTGTGGCA CCACGACACT GAATGGTCTG TGGCTGGATG ATGTTGTTTA TTGTCCGCGT
121 CATGTTATTT GTACCAGCGA AGATATGCTG AACCCGAATT ATGAAGATCT GCTGATTCGC
181 AAAAGCAACC ATAATTTTCT GGTTCAGGCA GGTAATGTTC AGCTGCGTGT TATTGGTCAT
241 AGCATGCAGA ATTGTGTGCT GAAACTGAAA GTTGATACCG CCAATCCGAA AACGCCGAAA
301 TATAAGTTTG TTCGTATTCA GCCTGGTCAG ACCTTTAGCG TTCTGGCATG TTATAATGGT
361 AGCCCGAGCG GTGTTTATCA GTGTGCAATG CGTCCGAATT TTACCATTAA AGGCAGCTTT
421 CTGAATGGTA GCTGTGGTAG CGTTGGTTTC AACATTGATT ATGATTGCGT GAGCTTCTGC
481 TATATGCATC ATATGGAACT GCCGACCGGT GTTCATGCAG GCACCGATCT GGAAGGTAAC
541 TTTTATGGTC CGTTTGTTGA TCGTCAGACC GCACAGGCAG CAGGTACAGA TACCACCATT
601 ACCGTTAATG TTCTGGCCTG GCTGTATGCA GCAGTTATTA ATGGTGATCG CTGGTTTCTG
661 AATCGTTTTA CAACAACCCT GAACGATTTT AATCTGGTGG CCATGAAATA TAACTATGAA
721 CCGCTGACAC AGGATCATGT TGATATTCTG GGTCCGCTGA GCGCACAGAC CGGTATTGCA
781 GTTCTGGATA TGTGTGCAAG CCTGAAAGAA CTGTTACAGA ATGGTATGAA TGGTCGTACA
841 ATTCTGGGTA GCGCACTGCT GGAAGATGAA TTCACCCCGT TTGATGTTGT GCGTCAGTGT
901 AGCGGTGTTA CCTTTCAGAG CGGT
<210> SEQ ID NO:2
<211> 6981 п.н.
<212> DNA
<213> Artificial Sequence
<220>
<223> Нуклеотидная последовательность рекомбинантной плазмиды pET-GST-3CL
<400> 2
1 TGGCGAATGG GACGCGCCCT GTAGCGGCGC ATTAAGCGCG GCGGGTGTGG TGGTTACGCG
61 CAGCGTGACC GCTACACTTG CCAGCGCCCT AGCGCCCGCT CCTTTCGCTT TCTTCCCTTC
121 CTTTCTCGCC ACGTTCGCCG GCTTTCCCCG TCAAGCTCTA AATCGGGGGC TCCCTTTAGG
181 GTTCCGATTT AGTGCTTTAC GGCACCTCGA CCCCAAAAAA CTTGATTAGG GTGATGGTTC
241 ACGTAGTGGG CCATCGCCCT GATAGACGGT TTTTCGCCCT TTGACGTTGG AGTCCACGTT
301 CTTTAATAGT GGACTCTTGT TCCAAACTGG AACAACACTC AACCCTATCT CGGTCTATTC
361 TTTTGATTTA TAAGGGATTT TGCCGATTTC GGCCTATTGG TTAAAAAATG AGCTGATTTA
421 ACAAAAATTT AACGCGAATT TTAACAAAAT ATTAACGTTT ACAATTTCAG GTGGCACTTT
481 TCGGGGAAAT GTGCGCGGAA CCCCTATTTG TTTATTTTTC TAAATACATT CAAATATGTA
541 TCCGCTCATG AGACAATAAC CCTGATAAAT GCTTCAATAA TATTGAAAAA GGAAGAGTAT
601 GAGTATTCAA CATTTCCGTG TCGCCCTTAT TCCCTTTTTT GCGGCATTTT GCCTTCCTGT
661 TTTTGCTCAC CCAGAAACGC TGGTGAAAGT AAAAGATGCT GAAGATCAGT TGGGTGCACG
721 AGTGGGTTAC ATCGAACTGG ATCTCAACAG CGGTAAGATC CTTGAGAGTT TTCGCCCCGA
781 AGAACGTTTT CCAATGATGA GCACTTTTAA AGTTCTGCTA TGTGGCGCGG TATTATCCCG
841 TATTGACGCC GGGCAAGAGC AACTCGGTCG CCGCATACAC TATTCTCAGA ATGACTTGGT
901 TGAGTACTCA CCAGTCACAG AAAAGCATCT TACGGATGGC ATGACAGTAA GAGAATTATG
961 CAGTGCTGCC ATAACCATGA GTGATAACAC TGCGGCCAAC TTACTTCTGA CAACGATCGG
1021 AGGACCGAAG GAGCTAACCG CTTTTTTGCA CAACATGGGG GATCATGTAA CTCGCCTTGA
1081 TCGTTGGGAA CCGGAGCTGA ATGAAGCCAT ACCAAACGAC GAGCGTGACA CCACGATGCC
1141 TGCAGCAATG GCAACAACGT TGCGCAAACT ATTAACTGGC GAACTACTTA CTCTAGCTTC
1201 CCGGCAACAA TTAATAGACT GGATGGAGGC GGATAAAGTT GCAGGACCAC TTCTGCGCTC
1261 GGCCCTTCCG GCTGGCTGGT TTATTGCTGA TAAATCTGGA GCCGGTGAGC GTGGGTCTCG
1321 CGGTATCATT GCAGCACTGG GGCCAGATGG TAAGCCCTCC CGTATCGTAG TTATCTACAC
1381 GACGGGGAGT CAGGCAACTA TGGATGAACG AAATAGACAG ATCGCTGAGA TAGGTGCCTC
1441 ACTGATTAAG CATTGGTAAC TGTCAGACCA AGTTTACTCA TATATACTTT AGATTGATTT
1501 AAAACTTCAT TTTTAATTTA AAAGGATCTA GGTGAAGATC CTTTTTGATA ATCTCATGAC
1561 CAAAATCCCT TAACGTGAGT TTTCGTTCCA CTGAGCGTCA GACCCCGTAG AAAAGATCAA
1621 AGGATCTTCT TGAGATCCTT TTTTTCTGCG CGTAATCTGC TGCTTGCAAA CAAAAAAACC
1681 ACCGCTACCA GCGGTGGTTT GTTTGCCGGA TCAAGAGCTA CCAACTCTTT TTCCGAAGGT
1741 AACTGGCTTC AGCAGAGCGC AGATACCAAA TACTGTCCTT CTAGTGTAGC CGTAGTTAGG
1801 CCACCACTTC AAGAACTCTG TAGCACCGCC TACATACCTC GCTCTGCTAA TCCTGTTACC
1861 AGTGGCTGCT GCCAGTGGCG ATAAGTCGTG TCTTACCGGG TTGGACTCAA GACGATAGTT
1921 ACCGGATAAG GCGCAGCGGT CGGGCTGAAC GGGGGGTTCG TGCACACAGC CCAGCTTGGA
1981 GCGAACGACC TACACCGAAC TGAGATACCT ACAGCGTGAG CTATGAGAAA GCGCCACGCT
2041 TCCCGAAGGG AGAAAGGCGG ACAGGTATCC GGTAAGCGGC AGGGTCGGAA CAGGAGAGCG
2101 CACGAGGGAG CTTCCAGGGG GAAACGCCTG GTATCTTTAT AGTCCTGTCG GGTTTCGCCA
2161 CCTCTGACTT GAGCGTCGAT TTTTGTGATG CTCGTCAGGG GGGCGGAGCC TATGGAAAAA
2221 CGCCAGCAAC GCGGCCTTTT TACGGTTCCT GGCCTTTTGC TGGCCTTTTG CTCACATGTT
2281 CTTTCCTGCG TTATCCCCTG ATTCTGTGGA TAACCGTATT ACCGCCTTTG AGTGAGCTGA
2341 TACCGCTCGC CGCAGCCGAA CGACCGAGCG CAGCGAGTCA GTGAGCGAGG AAGCGGAAGA
2401 GCGCCTGATG CGGTATTTTC TCCTTACGCA TCTGTGCGGT ATTTCACACC GCATATATGG
2461 TGCACTCTCA GTACAATCTG CTCTGATGCC GCATAGTTAA GCCAGTATAC ACTCCGCTAT
2521 CGCTACGTGA CTGGGTCATG GCTGCGCCCC GACACCCGCC AACACCCGCT GACGCGCCCT
2581 GACGGGCTTG TCTGCTCCCG GCATCCGCTT ACAGACAAGC TGTGACCGTC TCCGGGAGCT
2641 GCATGTGTCA GAGGTTTTCA CCGTCATCAC CGAAACGCGC GAGGCAGCTG CGGTAAAGCT
2701 CATCAGCGTG GTCGTGAAGC GATTCACAGA TGTCTGCCTG TTCATCCGCG TCCAGCTCGT
2761 TGAGTTTCTC CAGAAGCGTT AATGTCTGGC TTCTGATAAA GCGGGCCATG TTAAGGGCGG
2821 TTTTTTCCTG TTTGGTCACT GATGCCTCCG TGTAAGGGGG ATTTCTGTTC ATGGGGGTAA
2881 TGATACCGAT GAAACGAGAG AGGATGCTCA CGATACGGGT TACTGATGAT GAACATGCCC
2941 GGTTACTGGA ACGTTGTGAG GGTAAACAAC TGGCGGTATG GATGCGGCGG GACCAGAGAA
3001 AAATCACTCA GGGTCAATGC CAGCGCTTCG TTAATACAGA TGTAGGTGTT CCACAGGGTA
3061 GCCAGCAGCA TCCTGCGATG CAGATCCGGA ACATAATGGT GCAGGGCGCT GACTTCCGCG
3121 TTTCCAGACT TTACGAAACA CGGAAACCGA AGACCATTCA TGTTGTTGCT CAGGTCGCAG
3181 ACGTTTTGCA GCAGCAGTCG CTTCACGTTC GCTCGCGTAT CGGTGATTCA TTCTGCTAAC
3241 CAGTAAGGCA ACCCCGCCAG CCTAGCCGGG TCCTCAACGA CAGGAGCACG ATCATGCGCA
3301 CCCGTGGGGC CGCCATGCCG GCGATAATGG CCTGCTTCTC GCCGAAACGT TTGGTGGCGG
3361 GACCAGTGAC GAAGGCTTGA GCGAGGGCGT GCAAGATTCC GAATACCGCA AGCGACAGGC
3421 CGATCATCGT CGCGCTCCAG CGAAAGCGGT CCTCGCCGAA AATGACCCAG AGCGCTGCCG
3481 GCACCTGTCC TACGAGTTGC ATGATAAAGA AGACAGTCAT AAGTGCGGCG ACGATAGTCA
3541 TGCCCCGCGC CCACCGGAAG GAGCTGACTG GGTTGAAGGC TCTCAAGGGC ATCGGTCGAG
3601 ATCCCGGTGC CTAATGAGTG AGCTAACTTA CATTAATTGC GTTGCGCTCA CTGCCCGCTT
3661 TCCAGTCGGG AAACCTGTCG TGCCAGCTGC ATTAATGAAT CGGCCAACGC GCGGGGAGAG
3721 GCGGTTTGCG TATTGGGCGC CAGGGTGGTT TTTCTTTTCA CCAGTGAGAC GGGCAACAGC
3781 TGATTGCCCT TCACCGCCTG GCCCTGAGAG AGTTGCAGCA AGCGGTCCAC GCTGGTTTGC
3841 CCCAGCAGGC GAAAATCCTG TTTGATGGTG GTTAACGGCG GGATATAACA TGAGCTGTCT
3901 TCGGTATCGT CGTATCCCAC TACCGAGATA TCCGCACCAA CGCGCAGCCC GGACTCGGTA
3961 ATGGCGCGCA TTGCGCCCAG CGCCATCTGA TCGTTGGCAA CCAGCATCGC AGTGGGAACG
4021 ATGCCCTCAT TCAGCATTTG CATGGTTTGT TGAAAACCGG ACATGGCACT CCAGTCGCCT
4081 TCCCGTTCCG CTATCGGCTG AATTTGATTG CGAGTGAGAT ATTTATGCCA GCCAGCCAGA
4141 CGCAGACGCG CCGAGACAGA ACTTAATGGG CCCGCTAACA GCGCGATTTG CTGGTGACCC
4201 AATGCGACCA GATGCTCCAC GCCCAGTCGC GTACCGTCTT CATGGGAGAA AATAATACTG
4261 TTGATGGGTG TCTGGTCAGA GACATCAAGA AATAACGCCG GAACATTAGT GCAGGCAGCT
4321 TCCACAGCAA TGGCATCCTG GTCATCCAGC GGATAGTTAA TGATCAGCCC ACTGACGCGT
4381 TGCGCGAGAA GATTGTGCAC CGCCGCTTTA CAGGCTTCGA CGCCGCTTCG TTCTACCATC
4441 GACACCACCA CGCTGGCACC CAGTTGATCG GCGCGAGATT TAATCGCCGC GACAATTTGC
4501 GACGGCGCGT GCAGGGCCAG ACTGGAGGTG GCAACGCCAA TCAGCAACGA CTGTTTGCCC
4561 GCCAGTTGTT GTGCCACGCG GTTGGGAATG TAATTCAGCT CCGCCATCGC CGCTTCCACT
4621 TTTTCCCGCG TTTTCGCAGA AACGTGGCTG GCCTGGTTCA CCACGCGGGA AACGGTCTGA
4681 TAAGAGACAC CGGCATACTC TGCGACATCG TATAACGTTA CTGGTTTCAC ATTCACCACC
4741 CTGAATTGAC TCTCTTCCGG GCGCTATCAT GCCATACCGC GAAAGGTTTT GCGCCATTCG
4801 ATGGTGTCCG GGATCTCGAC GCTCTCCCTT ATGCGACTCC TGCATTAGGA AGCAGCCCAG
4861 TAGTAGGTTG AGGCCGTTGA GCACCGCCGC CGCAAGGAAT GGTGCATGCA AGGAGATGGC
4921 GCCCAACAGT CCCCCGGCCA CGGGGCCTGC CACCATACCC ACGCCGAAAC AAGCGCTCAT
4981 GAGCCCGAAG TGGCGAGCCC GATCTTCCCC ATCGGTGATG TCGGCGATAT AGGCGCCAGC
5041 AACCGCACCT GTGGCGCCGG TGATGCCGGC CACGATGCGT CCGGCGTAGA GGATCGAGAT
5101 CTCGATCCCG CGAAATTAAT ACGACTCACT ATAGGGGAAT TGTGAGCGGA TAACAATTCC
5161 CCTCTAGAAA TAATTTTGTT TAACTTTAAG AAGGAGATAT ACATATGTCC CCTATACTAG
5221 GTTATTGGAA AATTAAGGGC CTTGTGCAAC CCACTCGACT TCTTTTGGAA TATCTTGAAG
5281 AAAAATATGA AGAGCATTTG TATGAGCGCG ATGAAGGTGA TAAATGGCGA AACAAAAAGT
5341 TTGAATTGGG TTTGGAGTTT CCCAATCTTC CTTATTATAT TGATGGTGAT GTTAAATTAA
5401 CACAGTCTAT GGCCATCATA CGTTATATAG CTGACAAGCA CAACATGTTG GGTGGTTGTC
5461 CAAAAGAGCG TGCAGAGATT TCAATGCTTG AAGGAGCGGT TTTGGATATT AGATACGGTG
5521 TTTCGAGAAT TGCATATAGT AAAGACTTTG AAACTCTCAA AGTTGATTTT CTTAGCAAGC
5581 TACCTGAAAT GCTGAAAATG TTCGAAGATC GTTTATGTCA TAAAACATAT TTAAATGGTG
5641 ATCATGTAAC CCATCCTGAC TTCATGTTGT ATGACGCTCT TGATGTTGTT TTATACATGG
5701 ACCCAATGTG CCTGGATGCG TTCCCAAAAT TAGTTTGTTT TAAAAAACGT ATTGAAGCTA
5761 TCCCACAAAT TGATAAGTAC TTGAAATCCA GCAAGTATAT AGCATGGCCT TTGCAGGGCT
5821 GGCAAGCCAC GTTTGGTGGT GGCGACCATC CTCCAAAAGG ATCCACCTCA GCTGTTTTGC
5881 AGAGCGGTTT TCGTAAAATG GCATTTCCGA GCGGTAAAGT TGAAGGTTGT ATGGTTCAGG
5941 TTACCTGTGG CACCACGACA CTGAATGGTC TGTGGCTGGA TGATGTTGTT TATTGTCCGC
6001 GTCATGTTAT TTGTACCAGC GAAGATATGC TGAACCCGAA TTATGAAGAT CTGCTGATTC
6061 GCAAAAGCAA CCATAATTTT CTGGTTCAGG CAGGTAATGT TCAGCTGCGT GTTATTGGTC
6121 ATAGCATGCA GAATTGTGTG CTGAAACTGA AAGTTGATAC CGCCAATCCG AAAACGCCGA
6181 AATATAAGTT TGTTCGTATT CAGCCTGGTC AGACCTTTAG CGTTCTGGCA TGTTATAATG
6241 GTAGCCCGAG CGGTGTTTAT CAGTGTGCAA TGCGTCCGAA TTTTACCATT AAAGGCAGCT
6301 TTCTGAATGG TAGCTGTGGT AGCGTTGGTT TCAACATTGA TTATGATTGC GTGAGCTTCT
6361 GCTATATGCA TCATATGGAA CTGCCGACCG GTGTTCATGC AGGCACCGAT CTGGAAGGTA
6421 ACTTTTATGG TCCGTTTGTT GATCGTCAGA CCGCACAGGC AGCAGGTACA GATACCACCA
6481 TTACCGTTAA TGTTCTGGCC TGGCTGTATG CAGCAGTTAT TAATGGTGAT CGCTGGTTTC
6541 TGAATCGTTT TACAACAACC CTGAACGATT TTAATCTGGT GGCCATGAAA TATAACTATG
6601 AACCGCTGAC ACAGGATCAT GTTGATATTC TGGGTCCGCT GAGCGCACAG ACCGGTATTG
6661 CAGTTCTGGA TATGTGTGCA AGCCTGAAAG AACTGTTACA GAATGGTATG AATGGTCGTA
6721 CAATTCTGGG TAGCGCACTG CTGGAAGATG AATTCACCCC GTTTGATGTT GTGCGTCAGT
6781 GTAGCGGTGT TACCTTTCAG AGCGGTTAAG CGGCCGCACT CGAGCACCAC CACCACCACC
6841 ACTGAGATCC GGCTGCTAAC AAAGCCCGAA AGGAAGCTGA GTTGGCTGCT GCCACCGCTG
6901 AGCAATAACT AGCATAACCC CTTGGGGCCT CTAAACGGGT CTTGAGGGGT TTTTTGCTGA
6961 AAGGAGGAAC TATATCCGGA T 6981
<210> SEQ ID NO:3
<211> 308 а.о.
<212> DNA
<213> Artificial Sequence
<220>
<223> Аминокислотная последовательность рекомбинантного белка 3CL
<400> 3
1 SGFRKMAFPS GKVEGCMVQV TCGTTTLNGL WLDDVVYCPR HVICTSEDML NPNYEDLLIR
61 KSNHNFLVQA GNVQLRVIGH SMQNCVLKLK VDTANPKTPK YKFVRIQPGQ TFSVLACYNG
121 SPSGVYQCAM RPNFTIKGSF LNGSCGSVGF NIDYDCVSFC YMHHMELPTG VHAGTDLEGN
181 FYGPFVDRQT AQAAGTDTTI TVNVLAWLYA AVINGDRWFL NRFTTTLNDF NLVAMKYNYE
241 PLTQDHVDIL GPLSAQTGIA VLDMCASLKE LLQNGMNGRT ILGSALLEDE FTPFDVVRQC
301 SGVTFQSG
<---

Изобретение относится к биотехнологии. Описана рекомбинантная плазмида pET-GST-3CL, обеспечивающая синтез и транспорт химерной протеазы 3CL SARS-CoV-2 в периплазматическое пространство клеток E. coli в растворимой форме, имеющая нуклеотидную последовательность SEQ ID NO: 2 размером 6981 п.н., и содержащая в соответствии с физической и генетической картой, представленной на фиг. 1, следующие элементы: - AmpR promoter, имеющий координаты с 494 п.н. по 598 п.н.; - генетический маркер, определяющий устойчивость к ампициллину клеток бактерии E. coli, трансформированных рекомбинантной плазмидой AmpR, и имеющий координаты с 599 п.н. по 1459 п.н.; - участок начала репликации ori, имеющий координаты с 1630 по 2218 п.н.; - lacI - ДНК-связывающий белок, который ингибирует экспрессию генов E. coli, обеспечивает экспрессию целевого рекомбинантного белка 3CL после добавления индуктора изопропил-β-D-1-тиогалактопиранозида (IPTG) и имеет координаты с 3648 по 4730 п.н.; - lacI promoter, имеющий координаты с 4731 по 4808 п.н.; - промотор фага Т7, имеющий координаты с 5117 по 5135 п.н. и обеспечивающий экспрессию последовательности гена GST-3CL в клетках E. coli; - lac operator, имеющий координаты с 5136 по 5160 п.н.; - RBS - сайт связывания рибосомы, имеющий координаты с 5191 по 5196 п.н.; - ген GST - глутатион-S-трансфераза из Schistosoma japonicum, имеющий координаты с 5205 по 5857 п.н.; - Pep - участок, кодирующий пептид GSTSAVLQ, и имеющий координаты с 5859 по 5882 п.н.; - химерный ген 3CL SARS-CoV-2 с нуклеотидной последовательностью SEQ ID NO: 1, имеющий координаты с 5883 по 6806 п.н. и кодирующий синтез белка протеазы 3CL с аминокислотной последовательностью SEQ ID NO: 3; - Т7 terminator, имеющий координаты с 6909 по 6956 п.н. Изобретение расширяет арсенал получения белка 3CL. 3 ил., 1 табл., 5 пр.
Рекомбинантная плазмида pET-GST-3CL, обеспечивающая синтез и транспорт химерной протеазы 3CL SARS-CoV-2 в периплазматическое пространство клеток E. coli в растворимой форме, имеющая нуклеотидную последовательность SEQ ID NO: 2 размером 6981 п.н., и содержащая в соответствии с физической и генетической картой, представленной на фиг. 1, следующие элементы: - AmpR promoter, имеющий координаты с 494 п.н. по 598 п.н.; - генетический маркер, определяющий устойчивость к ампициллину клеток бактерии E. coli, трансформированных рекомбинантной плазмидой AmpR, и имеющий координаты с 599 п.н. по 1459 п.н.; - участок начала репликации ori, имеющий координаты с 1630 по 2218 п.н.; - lacI - ДНК-связывающий белок, который ингибирует экспрессию генов E. coli, обеспечивает экспрессию целевого рекомбинантного белка 3CL после добавления индуктора изопропил-β-D-1-тиогалактопиранозида (IPTG) и имеет координаты с 3648 по 4730 п.н.; - lacI promoter, имеющий координаты с 4731 по 4808 п.н.; - промотор фага Т7, имеющий координаты с 5117 по 5135 п.н. и обеспечивающий экспрессию последовательности гена GST-3CL в клетках E. coli; - lac operator, имеющий координаты с 5136 по 5160 п.н.; - RBS - сайт связывания рибосомы, имеющий координаты с 5191 по 5196 п.н.; - ген GST - глутатион-S-трансфераза из Schistosoma japonicum, имеющий координаты с 5205 по 5857 п.н.; - Pep - участок, кодирующий пептид GSTSAVLQ, и имеющий координаты с 5859 по 5882 п.н.; - химерный ген 3CL SARS-CoV-2 с нуклеотидной последовательностью SEQ ID NO: 1, имеющий координаты с 5883 по 6806 п.н. и кодирующий синтез белка протеазы 3CL с аминокислотной последовательностью SEQ ID NO: 3; - Т7 terminator, имеющий координаты с 6909 по 6956 п.н.
| WO 2021262734 A2, 30.12.2021 | |||
| WO 2021236550 A1, 25.11.2021 | |||
| CN 111979214 A, 24.11.2020 | |||
| RESNICK SJ, IKETANI S и др., Inhibitors of Coronavirus 3CL Proteases Protect Cells from Protease-Mediated Cytotoxicity, J Virol | |||
| Способ регенерирования сульфо-кислот, употребленных при гидролизе жиров | 1924 |
|
SU2021A1 |
| RU 2020123357 A, 14.01.2022. | |||
Авторы
Даты
2023-03-16—Публикация
2022-03-16—Подача