[1] Настоящей заявке испрашивается приоритет по предварительной патентной заявке США с серийным номером №62/527764, поданной 30 июня 2017 года, и предварительной патентной заявке США с серийным номером №62/528846, поданной 5 июля 2017 года, обе из которых включены в настоящее описание в качестве ссылок в полном объеме для любых целей.
ОБЛАСТЬ ИЗОБРЕТЕНИЯ
[2] Настоящее изобретение относится сконструированным вариантам РНК-полимеразы и к их композициям, содержащим эти варианты. Кроме того, настоящее изобретение относится к сконструированным вариантам РНК-полимеразы T7 и к композициям, содержащим эти варианты. Эти варианты изменены для селективного включения аналога кэпа m7G(5')ppp(5')m7G относительно GTP при инициации транскрипции in vitro. Настоящее изобретение также относится к способам применения вариантов, описанных в настоящем описании. Кроме того, настоящее изобретение относится к применению композиций, описанных в настоящем описании.
УКАЗАНИЕ НА СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ, ТАБЛИЦУ ИЛИ КОМПЬЮТЕРНУЮ ПРОГРАММУ
[3] Официальная копия списка последовательностей предоставлена одновременно с описанием в качестве текстового файла в формате ASCII через EFS-Web, с названием файла "CX8-168CUSP1_ST25.txt", датой создания 5 июля 2017 года и размером 184,320 байт. Список последовательностей, предоставленный через EFS-Web, является частью описания и включен в настоящее описание в качестве ссылки в полном объеме.
УРОВЕНЬ ТЕХНИКИ, К КОТОРОМУ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
[4] РНК-полимераза T7 (E.C. 2.7.7.6) представляет собой мономерную кодируемую бактериофагом DNA-направляемую РНК-полимеразу, которая катализирует образование РНК в направлении от 5' к 3'. В процессе инициации транскрипции РНК-полимераза T7 распознает конкретную промоторную последовательность (т.е. промотор T7). Встречающаяся в природе РНК-полимераза T7 содержит 883 аминокислоты. Она является высокогомологичной РНК-полимеразе T3 и в некоторой степени гомологичной РНК-полимеразе SP6. T7 содержит несколько доменов, включая N-концевой домен, "большой палец", "ладонь" и "пальцы" (см., например, Sousa and Mukherjee, Prog. Nucl. Acid Res. Mol. Biol., 73:1-41 [2003] и патент США №9193959). Конформация N-концевого домена меняется между фазами инициации и элонгации функционирующего фермента.
[5] Описано клонирование и экспрессия гена, кодирующего РНК-полимеразу T7 (см., например, патент США №4952496). Вследствие ее специфичности к промотору и высокой активности РНК-полимеразы, T7 используется для различных применений. Также она является пригодной для экспрессии на высоком уровне рекомбинантных генов в E. coli (См., Studier and Moffat, J. Mol. Biol., 18:113-130 [1986]). T7 также используется в различных способах амплификации нуклеиновых кислот, включая те, которые используются в диагностических способах. Поскольку стабильность и термостабильность часто являются важными факторами, которые должны учитываться при разработке компонентов диагностических способов, описана работа по повышению термостабильности и стабильности T7 (см., например, патенты США №9193959, 8551752 и 7507567 и т.д.). Тем не менее в данной области остается потребность в вариантах ферментов T7, которые демонстрируют улучшенные свойства по сравнению со встречающимся в природе ферментом.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
[6] Настоящее изобретение относится сконструированным вариантам РНК-полимеразы T7 и их композициям. Эти варианты изменены для селективного включения симметричного кэпированного аналога GTP относительно GTP при инициации транскрипции in vitro. Настоящее изобретение также относится к способам применения вариантов, описанных в настоящем описании. Кроме того, настоящее изобретение относится к применению композиций, описанных в настоящем описании.
[7] Настоящее изобретение относится сконструированным РНК-полимеразам, содержащим полипептидные последовательности, обладающие по меньшей мере 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичностью последовательности с эталонной последовательностью SEQ ID NO: 4 и/или 15, или их функциональным фрагментам, где сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен в полипептидных последовательностях и где аминокислотные положения полипептидных последовательностей пронумерованы относительно SEQ ID NO: 4 или 15.
[8] В некоторых вариантах осуществления сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из 32/357, 49/642, 97/357, 136, 137, 160/643, 167/514, 250, 302/513, 314/401, 357, 392, 393, 394, 397, 401, 404, 444, 446, 478, 513, 514, 582, 635, 636, 637, 639, 642, 643, 645, 653, 656, 660, 660/806, 661 и 664, и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 4. В некоторых других вариантах осуществления сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из 32V/357I, 49G/642L, 97D/357G, 136E/I, 137W, 160L/643S, 167N/514L, 250D, 302V/513G, 314C/401V, 357G/K/L/M/N/Q/R/S/T/V/W, 392D, 393L/Y, 394A/L/R, 397F/M/Q/W, 401A/I/L/S/V, 404E/Y, 444F/H/I/V, 446W/Y, 478F/M/W, 513C/F/K/L/R/T/W, 514F/I/L/Y, 582N, 635W, 636L, 637G/P/S, 639H, 642L, 643A, 645V, 653C, 656F/W, 660A/C/M/S/T/W, 660N/806Y, 661E/Y и 664W, и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 4. В некоторых дополнительных вариантах осуществления сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из A32V/E357I, E49G/M642L, E97D/E357G, A136E/I, D137W, R160L/T643S, K167N/S514L, T250D, A302V/D513G, R314C/R401V, E357G/K/L/M/N/Q/R/S/T/V/W, Y392D, R393L/Y, K394A/L/R, A397F/M/Q/W, R401A/I/L/S/V, S404E/Y, N444F/H/I/V, M446W/Y, D478F/M/W, D513C/F/K/L/R/T/W, S514F/I/L/Y, A582N, S635W, V636L, T637G/P/S, R639H, M642L, T643A, A645V, F653C, Q656F/W, D660A/C/M/S/T/W, D660N/H806Y, T661E/Y и P664W, и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 4.
[9] В некоторых вариантах осуществления сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из 397/513/635, 397/513/635/660, 513/660/664, 513/635/660, 513/635/664, 513/660/664 и 660/664, и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 4. В некоторых других вариантах осуществления сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из 397F/513W/635W, 397F/513Y/635W, 397W/513Y/635W, 397W/513W/635W, 397W/513Y/635W/660F, 397W/513W/635W/660W, 397Y/513W/635W/660F, 475V/513W/635W/660Y, 513F/660W/664Y, 513W/635W/660F, 513W/635W/664W, 513Y/635W/660F, 513Y/635W/660Y, 513Y/660W/664W и 660Y/664W, и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 4. В некоторых дополнительных вариантах осуществления сконструированные РНК полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из A397F/D513W/S635W, A397F/D513Y/S635W, A397W/D513Y/S635W, A397W/D513W/S635W, A397W/D513Y/S635W/D660F, A397W/D513W/S635W/D660W, A397Y/D513W/S635W/D660F, A475V/D513W/S635W/D660Y, D513F/D660W/P664Y, D513W/S635W/D660F, D513W/S635W/P664W, D513Y/S635W/D660F, D513Y/S635W/D660Y, D513Y/D660W/P664W и D660Y/P664W, и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 4.
[10] В некоторых вариантах осуществления сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из 397, 397/513, 397/513/635, 397/513/635, 397/513/635/656, 397/513/635/656/660, 397/513/635/656/660/664, 397/513/635/656/664, 397/513/635/660, 397/513/635/660/664, 397/513/635/664, 397/513/656/660, 397/513/660, 397/513/660/664, 397/513/664, 397/513, 397/635, 397/635/656/660/664, 397/635/656/664, 397/635/660, 397/635/664, 397/635/664/850, 397/660, 397/664, 397/660/664, 397/837, 399/635/660, 475/513/635/660, 513, 513/635, 513/635/656, 513/635/656/660, 513/635/656/664, 513/635/660, 513/635/660/664, 513/635/664, 513/656/660, 513/656/664, 513/660, 513/660/664, 513/664, 635, 635/656, 635/656/664, 635/660, 635/660/664, 635/664, 656/660/664, 658, 660, 660/664 и 664, и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 4. В некоторых других вариантах осуществления сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из 397F, 397F/513F, 397F/513F/635F, 397F/513F/635W, 397F/513F/635W/660W, 397F/513W/635W, 397F/513W/635W/656F/664F, 397F/513W/664W, 397F/513Y, 397F/513Y/635W, 397F/513Y/635W/656W, 397F/513Y/635W/664F, 37F/513Y/656H/660W, 397F/635F/660W, 397F/635W, 397F/660W, 397F/664W, 397W, 397W/513F, 397W/513F/635F/656F/660F/664W, 397W/513F/635F/660W, 397W/513F/635W, 397W/513F/660F, 397W/513F/660W, 397W/513F/660Y/664F, 397W/513W, 397W/513W/635F, 397W/513W/635F/656W/660F, 397W/513W/635W, 397W/513W/635W/656Y, 397W/513W/635W/660W, 397W/513W/635W/660W/664Y, 397W/513W/635W/660Y/664Y, 397W/513W/660W, 397W/513W/660W/664Y, 397W/513W/664F, 397W/513W/664W, 397W/513Y/635F, 397W/513Y/635W/656Y/660W/664W, 397W/513Y/635W, 397W/513Y/635W/656Y/664Y, 397W/513Y/635W/660F, 397W/635F, 397W/635F/664F, 397W/635W, 397W/635W/656F/664F, 397W/635W/656F/664W, 397W/635W/656F/664Y, 397W/635W/660W, 397W/635W/660Y, 397W/635W/664F, 397W/635W/664W/850T, 397W/660F/664F, 397W/660F/664Y, 397W/660W, 397W/837K, 397Y, 397Y/513F/635W/656F/660W/664F, 397Y/513F/635W/664W, 397Y/513W/635F/664Y, 397Y/513W/635W/660F, 397Y/513W/656W/660W, 397Y/513Y, 397Y/513Y/635W, 397Y/635F, 397Y/635F/660W, 397Y/635W, 397Y/635W/656F/660Y/664W, 397Y/635W/660F, 397Y/660F/664W, 397Y/664F, 399E/635F/660W, 475V/513W/635W/660Y, 513F, 513F/635F, 513F/635W, 513F/635W/656W, 513F/635W/664W, 513F/660W, 513F/660W/664F, 513F/660W/664Y, 513W, 513W/635F, 513W/635W, 513Y/635F/660F/664Y, 513W/635W/656W/660F, 513W/635W/660F, 513W/635W/664W, 513W/656W/664W, 513W/656Y/660W, 513W/660F, 513W/660W, 513Y/635F, 513Y/635F/664W, 513Y/635R/656F/664Y, 513Y/635W, 513Y/635W/660F, 513Y/635W/660Y, 513Y/635W/660Y/664F, 513Y/660W, 513Y/660W/664W, 513Y/660Y/664F, 513Y/664Y, 635F/656F/664Y, 635F/656Y/664W, 635F/660W/664F, 635W, 635W/656W, 635W/660F, 635W/660W, 635W/664W, 656W/660F/664Y, 656W/660W/664Y, 658P, 660F, 660F/664F, 660F/664Y, 660W/664F, 660Y/664W, 664F и 664W, и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 4. В некоторых дополнительных вариантах осуществления сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из A397F, A397F/D513F, A397F/D513F/S635F, A397F/D513F/S635W, A397F/D513F/S635W/D660W, A397F/D513W/S635W, A397F/D513W/S635W/Q656F/P664F, A397F/D513W/P664W, A397F/D513Y, A397F/D513Y/S635W, A397F/D513Y/S635W/Q656W, A397F/D513Y/S635W/P664F, A397F/D513Y/Q656H/D660W, A397F/S635F/D660W, A397F/S635W, A397F/D660W, A397F/P664W, A397W, A397W/D513F, A397W/D513F/S635F/Q656F/D660F/P664W, A397W/D513F/S635F/D660W, A397W/D513F/S635W, A397W/D513F/D660F, A397W/D513F/D660W, A397W/D513F/D660Y/P664F, A397W/D513W, A397W/D513W/S635F, A397W/D513W/S635F/Q656W/D660F, A397W/D513W/S635W, A397W/D513W/S635W/Q656Y, A397W/D513W/S635W/D660W, A397W/D513W/S635W/D660W/P664Y, A397W/D513W/S635W/D660Y/P664Y, A397W/D513W/D660W, A397W/D513W/P664F, A397W/D513W/D660W/P664Y, A397W/D513W/P664W, A397W/D513Y/S635F, A397W/D513Y/S635W/Q656Y/D660W/P664W, A397W/D513Y/S635W, A397W/D513Y/S635W/Q656Y/P664Y, A397W/D513Y/S635W/D660F, A397W/S635F, A397W/S635F/P664F, A397W/S635W, A397W/S635W/Q656F/P664F, A397W/S635W/Q656F/P664W, A397W/S635W/Q656F/P664Y, A397W/S635W/D660W, A397W/S635W/D660Y, A397W/S635W/P664F, A397W/S635W/P664W/A850T, A397W/D660F/P664F, A397W/D660F/P664Y, A397W/D660W, A397W/E837K, A397Y, A397Y/D513F/S635W/Q656F/D660W/P664F, A397Y/D513F/S635W/P664W, A397Y/D513W/S635F/P664Y, A397Y/D513W/S635W/D660F, A397Y/D513W/Q656W/D660W, A397Y/D513Y, A397Y/D513Y/S635W, A397Y/S635F, A397Y/S635F/D660W, A397Y/S635W, A397Y/S635W/Q656F/D660Y/P664W, A397Y/S635W/D660F, A397Y/D660F/P664W, A397Y/P664F, K399E/S635F/D660W, A475V/D513W/S635W/D660Y, D513F, D513F/S635F, D513F/S635W, D513F/S635W/Q656W, D513F/S635W/P664W, D513F/D660W, D513F/D660W/P664F, D513F/D660W/P664Y, D513W, D513W/S635F, D513W/S635W, D513Y/S635F/D660F/P664Y, D513W/S635W/Q656W/D660F, D513W/S635W/D660F, D513W/S635W/P664W, D513W/Q656W/P664W, D513W/Q656Y/D660W, D513W/D660F, D513W/D660W, D513Y/S635F, D513Y/S635F/P664W, D513Y/S635R/Q656F/P664Y, D513Y/S635W, D513Y/S635W/D660F, D513Y/S635W/D660Y, D513Y/S635W/D660Y/P664F, D513Y/D660W, D513Y/D660W/P664W, D513Y/D660Y/P664F, D513Y/P664Y, S635F/Q656F/P664Y, S635F/Q656Y/P664W, S635F/D660W/P664F, S635W, S635W/Q656W, S635W/D660F, S635W/D660W, S635W/P664W, Q656W/D660F/P664Y, Q656W/D660W/P664Y, L658P, D660F, D660F/P664F, D660F/P664Y, D660W/P664F, D660Y/P664W, P664F/W и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 4.
[11] В некоторых вариантах осуществления сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из 113/137/513, 136/357/404/514, 136/357/514, 136/394/404/446, 136/401, 136/401/404, 136/404/446, 136/404/514, 136/446, 136/514, 137, 137/401, 137/401/513, 137/401/513, 137/513, 137/513/621, 137/635, 137/656, 357/394/401/404/514, 357/394/446/514, 357/514, 394/446/514, 401/404, 401/404/514, 401/513/635, 401/635, 513/635, 513/635/656, 513/660, 635/656, 635/660 и 660, и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 15. В некоторых других вариантах осуществления сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из 113M/137W/513R,136E/357I/404Y/514I, 136I/357I/514F, 136I/357K/514F, 136I/394R/404Y/446W, 136I/401V, 136I/401V/404Y, 136E/404Y/446W, 136E/404Y/514F, 136I/446W, 136E/514F, 136I/514I, 137W, 137W/401I, 137W/401S, 137W/401S/513R, 137W/401S/513W, 137W/401V, 137W/513W, 137W/513R/621R, 137W/635W, 137W/656F, 357N/394R/446W/514I, 357R/394R/401V/404Y/514L, 357R/514F, 394R/446W/514I, 401S/513R/635W, 401S/635W, 401V/404Y, 401V/404Y/514L, 513L/635W, 513L/660W, 513R/635W/656F, 635W/656F, 635W/660T и 660S/T, и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 15. В некоторых дополнительных вариантах осуществления сконструированные РНК-полимеразы содержат по меньшей мере одну замену или набор замен, которые выбраны из L113M/D137W/D513R, A136E/E357I/S404Y/S514I, A136E/S404Y/M446W, A136E/S404Y/S514F, A136E/S514F, A136I/E357I/S514F, A136I/E357K/S514F, A136I/K394R/S404Y/M446W, A136I/R401V, A136I/R401V/S404Y, A136I/M446W, A136I/S514I, D137W, D137W/R401I, D137W/R401S, D137W/R401S/D513R, D137W/R401S/D513W, D137W/R401V, D137W/D513W, D137W/D513R/K621R, D137W/S635W, D137W/Q656F, E357N/K394R/M446W/S514I, E357R/K394R/R401V/S404Y/S514L, E357R/S514F, K394R/M446W/S514I, R401S/D513R/S635W, R401S/S635W, R401V/S404Y, R401V/S404Y/S514L, D513L/S635W, D513L/D660W, D513R/S635W/Q656F, S635W/Q656F, S635W/D660T и D660S/T, и/или любых их комбинаций, где положения аминокислот пронумерованы в соответствии с SEQ ID NO: 15.
[12] В некоторых дополнительных вариантах осуществления сконструированные РНК-полимеразы содержат полипептидные последовательности, по меньшей мере на 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичные последовательности по меньшей мере одного варианта сконструированной РНК-полимеразы, указанного в таблице 5.3, 5.4, 5.5 и/или 5.6. В некоторых дополнительных вариантах осуществления сконструированная РНК-полимераза представляет собой вариант сконструированной полимеразы, представленный в таблице 5.3, 5.4, 5.5 и/или 5.6.
[13] В следующих дополнительных вариантах осуществления сконструированная РНК-полимераза содержит полипептидную последовательность, которая по меньшей мере на 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентична последовательности по меньшей мере одного варианта сконструированной РНК-полимеразы, указанного в SEQ ID NO: 4, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37 и/или 39. В некоторых других вариантах осуществления сконструированная РНК-полимераза представляет собой вариант сконструированной полимеразы, указанный в SEQ ID NO: 4, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37 или 39.
[14] В некоторых других вариантах осуществления сконструированная полимераза имеет по меньшей мере одно улучшенное свойство по сравнению с РНК-полимеразой T7 дикого типа. В некоторых следующих вариантах осуществления сконструированная РНК-полимераза демонстрирует по меньшей мере одно улучшенное свойство, выбранное из увеличенной селективности в отношении аналога кэпа относительно GTP в ходе инициации транскрипции, увеличенной экспрессии белка, увеличенной стабильности в буфере для хранения и увеличенной стабильности в условиях реакции. В некоторых дополнительных вариантах осуществления сконструированная РНК-полимераза сохраняет выход РНК, точность транскрипции, термостабильность, экспрессию белка, стабильность при -20°C или стабильность в условиях реакции, эквивалентных РНК-полимеразе T7 дикого типа. В некоторых дополнительных вариантах осуществления сконструированная РНК-полимераза является очищенной. В некоторых других вариантах осуществления настоящее изобретение относится к композициям, содержащим по меньшей мере одну сконструированную РНК-полимеразу, описанную в настоящем описании.
[15] Настоящее изобретение также относится к полинуклеотидным последовательностям, кодирующим по меньшей мере одну сконструированную РНК-полимеразу, описанную в настоящем описании. В некоторых вариантах осуществления полинуклеотидные последовательности, кодирующие по меньшей мере одну сконструированную РНК-полимеразу, обладают по меньшей мере 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичностью последовательности с эталонной последовательностью SEQ ID NO: 4 и/или 15, или их функциональным фрагментом, где сконструированная РНК-полимераза содержит по меньшей мере одну замену по меньшей мере в одном или более положениях аминокислот. В некоторых следующих вариантах осуществления полинуклеотидные последовательности, кодирующие сконструированные РНК-полимеразы, описанные в настоящем описании, обладают по меньшей мере 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичностью последовательности с SEQ ID NO: 4, 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37 и/или 39, или их функциональным фрагментом. В некоторых следующих вариантах осуществления полинуклеотидные последовательности, кодирующие по меньшей мере одну сконструированную РНК-полимеразу, обладают по меньшей мере 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичностью последовательности с SEQ ID NO: 3, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36 и/или 38. В некоторых дополнительных вариантах осуществления полинуклеотидные последовательности функционально связаны с контрольной последовательностью. В некоторых других вариантах осуществления полинуклеотидные последовательности являются кодон-оптимизированными.
[16] Настоящее изобретение также относится к экспрессирующим векторам, содержащим по меньшей мере одну полинуклеотидную последовательность, описанную в настоящем описании. Кроме того, настоящее изобретение относится к клеткам-хозяевам, содержащим по меньшей мере один экспрессирующий вектор, описанный в настоящем описании. Настоящее изобретение также относится к способам продуцирования сконструированной РНК-полимеразы в клетке-хозяине, включающим культивирование клетки-хозяина, описанной в настоящем описании, в подходящих условиях культивирования, чтобы продуцировалась по меньшей мере одна сконструированная РНК-полимераза. В некоторых вариантах осуществления способы дополнительно включают выделение по меньшей мере одной сконструированной РНК-полимеразы из культуры и/или клетки-хозяина. В некоторых дополнительных вариантах осуществления способы дополнительно включают стадию очистки по меньшей мере одной сконструированной РНК-полимеразы.
[17] Настоящее изобретение также относится к способам продуцирования кэпированных РНК-транскриптов, включающим предоставление композиции, содержащей: i) по меньшей мере одну сконструированную РНК-полимеразу, описанную в настоящем описании, динуклеотидный аналог кэпа, и ii) ДНК-матрицу; воздействие на ДНК-матрицу композиции в таких условиях, чтобы сконструированная РНК-полимераза продуцировала кэпированный РНК-транскрипт. В некоторых вариантах осуществления способов, динуклеотидный аналог кэпа представляет собой альфа, гамма-бис(N7-метилгуанозин)трифосфат (m7G(5')ppp(5')m7G) или антиреверсный аналог кэпа 3'-O-Me-m7G(5')ppp(5')G. В некоторых других вариантах осуществления способов динуклеотидный аналог кэпа представляет собой альфа, гамма-бис(N7-метилгуанозин)трифосфат. В некоторых дополнительных вариантах осуществления способов в реакционную смесь добавляют неорганическую пирофосфатазу.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
[18] На фиг.1 представлена химическая структура "m7G(5')ppp(5')m7G", также обозначаемого как "альфа, гамма-бис(N7-метилгуанозин)трифосфат", "кэпированный GTP", "симметричный аналог кэпа" или "кэп".
ОПИСАНИЕ ИЗОБРЕТЕНИЯ
[19] Настоящее изобретение относится сконструированным вариантам РНК-полимеразы T7 и их композициям. Эти варианты изменены для селективного включения аналога кэпа m7G(5')ppp(5')m7G относительно GTP при инициации транскрипции in vitro. Настоящее изобретение также относится к способам применения вариантов, описанных в настоящем описании. Кроме того, настоящее изобретение относится к композициям, описанным в настоящем описании. Настоящее изобретение относится к композициям и способам для эффективного получения кэпированной РНК. Большинство мРНК-транскриптов эукариотических клеток и большинство мРНК-транскриптов вирусов эукариот, а также другие формы эукариотический РНК (например, малая ядреная РНК [мяРНК] и пре-микроРНК [пре-мкРНК]) блокированы или "кэпированы" на их 5'-концах (см., например, патент США №8846348, включенный в настоящее описание в качестве ссылки в полном объеме). Кэп представляет собой гуаниновый нуклеозид, который связан через его 5'-углерод с трифосфатной группой, которая связана с 5'-углеродом самого крайнего 5'-нуклеотида первичного мРНК-транскрипта. В большинстве эукариот азот в 7 положении гуанина в нуклеотиде кэпа является метилированным.
[20] 5'-кэпы этих молекул РНК играют важные роли в стабильности и процессинге РНК. Кэп играет очень важную роль в метаболизме мРНК и требуется для процессинга и созревания РНК-транскриптов в ядре, транспорта мРНК из ядра в цитоплазму, стабильности мРНК и эффективной трансляции мРНК в белок (см., например, Lewis et al., Eur. J. Biochem., 247:461-9 [1997].). Структура 5'-кэпа вовлечена в инициацию синтеза белка эукариотических и вирусных мРНК. 5'-кэп также обеспечивает резистентность к 5'-экзонуклеазной активности и его отсутствие приводит к быстрой деградации мРНК (см., например, патент США №8836348; и Furiichi et al., Nat., 266:235-9 [1997]). Вследствие преимуществ, обеспечиваемых кэпированием, эффективный синтез кэпированных РНК-транскриптов имеет большое значение для различных функций, включая, но не ограничиваясь ими, синтез белков in vitro и in vivo.
[21] Синтез in vitro кэпированной РНК практически можно проводить посредством одного из двух ферментативных путей. Кэпирующий фермент вируса коровьей оспы имеет две субъединицы и три вида ферментативной активности, которые присоединяют структуру 7-метилгуанозинового кэпа к 5'-фосфату транскрибируемой in vitro РНК (см., Mao and Shuman, J. Biol. Chem., 269:24472-9 [1994]).
[22] Альтернативным способом синтеза in vitro кэпированной РНК является встраивание структуры кэпа в ходе транскрипции in vitro с использованием аналога кэпа. Эти 7-метилгуанозин-содержащие динуклеотиды содержат 5'-5'-трифосфатную связь и включаются в участок инициации транскрипции, что приводит к кэпированному 7-метилгуанозином РНК-продукту. РНК-полимераза T7 естественным образом инициирует транскрипцию включением гуанозина напротив остатка цитозина на матрице. Для достижения высокой эффективности кэпирования аналог гуанозинового кэпа должен присутствовать в реакции транскрипции in vitro в высокой концентрации относительно GTP, с которым он конкурирует за включение в первое положение мРНК. Одним полезным признаком котранскрипционного кэпирования in vitro является то, что, поскольку встраивание кэпа достигается в ходе инициации, конечная последовательность или вторичная структура полноразмерной мРНК не влияет на степень кэпирования. Кроме того, этот процесс требует использования только одного фермента (РНК-полимераза) в одностадийной реакции. Использование химически синтезированного аналога кэпа также повышает адаптируемость к использованию альтернативных структур нуклеотидных кэпов, которые не являются хорошо распознаваемыми для метилирования кэпирующим ферментом коровьей оспы. Однако аналог кэпа является дорогостоящим относительно нуклеотидов и других компонентов реакции и должен присутствовать в реакционной смеси в высокой концентрации. Настоящее изобретение относится к вариантам РНК-полимеразы T7, которые являются селективными в отношении включении аналогов кэпа относительно GTP при инициации транскрипции для обеспечения эффективного кэпирования при использовании сниженной концентрации аналога кэпа, обеспечивающей более экономичный и масштабируемый процесс продуцирования кэпированной РНК.
[23] В некоторых вариантах осуществления изобретения неорганическая пирофосфатаза присутствует в ходе реакции транскрипции in vitro для деградации пирофосфата, ингибитора продукта транскрипции РНК-полимеразой, в ортофосфат.
Сокращенные обозначения и определения:
[24] Если не определено иначе, все технические и научные термины, используемые в настоящем описании, как правило обладают тем же значением, которое обычно подразумевает средний специалист в области, к которой настоящее изобретение относится. Как правило, номенклатура, используемая в настоящем описании, и лабораторные методики культивирования клеток, молекулярной генетики, микробиологии, биохимии, органической химии, аналитической химии и химии нуклеиновых кислот, описанные ниже, представляют собой номенклатуру и методики, хорошо известные и часто используемые в данной области. Такие способы хорошо известны и описаны в многочисленных пособиях и справочниках, хорошо известных специалистам в данной области. Для химического синтеза и химического анализа используются стандартные способы или их модификации. Все патенты, патентные заявки, статьи и публикации, упомянутые в настоящем описании, как выше, так и ниже, включены в настоящее описание в качестве ссылки в полном объеме.
[25] Хотя для применения настоящего изобретения на практике применимы любые подходящие способы и материалы, сходные или эквивалентные тем, что описаны в настоящем описании, некоторые способы и материалы описаны в настоящем описании. Следует понимать, что настоящее изобретение не ограничивается конкретной описанной методологией, протоколами и реагентами, поскольку они могут варьироваться в зависимости от контекста, в котором они используются специалистами в данной области. Таким образом, термины, определенные непосредственно ниже, более полно описаны посредством заявки в целом. Все патенты, патентные заявки, статьи и публикации, упомянутые в настоящем описании, как выше, так и ниже, включены в настоящее описание в качестве ссылок в полном объеме.
[26] Так же, как используют в рамках изобретения, форма единственного числа включает форму множественного числа упоминаемых объектов, если контекст явно не указывает на иное.
[27] Числовые диапазоны являются охватывающими числа, определяющие диапазон. Таким образом, каждый числовой диапазон, описанный в настоящем описании, охватывает каждый более узкий числовой диапазон, который входит в такой более широкий числовой диапазон, как если бы все такие более узкие числовые диапазоны были прямо указаны в настоящем описании. Также подразумевается, что каждое максимальное (или минимальное) числовое ограничение, описанное в настоящем описании, включает каждое более низкое (или более высокое) числовое ограничение, как если бы такие более низкие (или более высокие) числовые ограничения были прямо указаны в настоящем описании.
[28] Термин "приблизительно" означает допустимую погрешность конкретной величины. В некоторых случаях "приблизительно" означает в пределах 0,05%, 0,5%, 1,0% или 2,0% от данного числового диапазона. В некоторых случаях "приблизительно" означает в пределах 1, 2, 3 или 4 стандартных отклонений от данной величины.
[29] Более того, заголовки, приведенные в настоящем описании, не являются ограничением различных аспектов или вариантов осуществления изобретения, которые могут быть отнесены к заявке в целом.
[30] Таким образом, термины, определенные непосредственно ниже, более полно определяются рассмотрением заявки в целом. Тем не менее для облегчения понимания изобретения ряд терминов определен ниже.
[31] Если нет иных указаний, нуклеиновые кислоты приводятся слева направо в ориентации от 5' к 3'; аминокислотные последовательности приводятся слева направо в ориентации от амино к карбокси, соответственно.
[32] Как используют в рамках изобретения, термин "содержащий" и родственные ему термины используются в их включающем значении (т.е. эквивалентно термину "включающий" и его соответствующим родственным терминам).
[33] Номер "EC" относится к Номенклатуре фермента Комитета по номенклатуре Международного союза биохимии и молекулярной биологии (NC-IUBMB). Биохимическая классификация IUBMB представляет собой систему числовой классификации для ферментов на основе химических реакций, которые они катализируют.
[34] "ATCC" относится к Американской коллекции типовых культур, чье хранилище биологических материалов включает гены и штаммы.
[35] "NCBI" относится к Национальному центру биологической информации и базам данных, представленным в нем.
[36] Как используют в рамках изобретения, "РНК-полимераза T7" относится к мономерной кодируемой бактериофагом T7 ДНК-направляемой РНК-полимеразе, которая катализирует образование РНК в направлении от 5' к 3'.
[37] Как используют в рамках изобретения, термин "кэп" относится к гуаниновому нуклеозиду, который связан через его 5'-углерод с трифосфатной группой, которая в свою очередь связана с 5'-углеродов самого крайнего 5'-нуклеотида мРНК-транскрипта. В некоторых вариантах осуществления азот в положении 7 гуанина в кэпе является метилированным.
[38] Как используют в рамках изобретения, термины "кэпированная РНК", "5'-кэпированная РНК" и "кэпированная мРНК" относятся к РНК и мРНК, соответственно, которые содержат кэп.
[39] Как используют в рамках изобретения, "полинуклеотид" и "нуклеиновая кислота" относятся к двум или более нуклеозидам, которые ковалентно связаны друг с другом. Полинуклеотид может полностью состоять из рибонуклеотидов (т.е. РНК), полностью состоять из 2'-дезоксирибонуклеотидов (т.е. ДНК) или состоять из смесей рибо- и 2'-дезоксирибонуклеотидов. В то время как нуклеозиды, как правило, связаны между собой стандартными фосфодиэфирными связями, полинуклеотиды могут включать одну или более нестандартных связей. Полинуклеотид может быть одноцепочечным или двухцепочечным, или может включать как одноцепочечные области, так и двухцепочечные области. Более того, в то время как полинуклеотид обычно состоит из встречающихся в природе кодирующих нуклеиновых оснований (т.е. аденин, гуанин, урацил, тимин и цитозин), он может включать одно или более модифицированных и/или синтетических нуклеиновых оснований, например, таких как инозин, ксантин, гипоксантин и т.д. В некоторых вариантах осуществления такие модифицированные или синтетические нуклеиновые основания представляют собой нуклеиновые основания, кодирующие аминокислотные последовательности.
[40] "Белок", "полипептид" и "пептид" используются в настоящем описании взаимозаменяемо для обозначения полимера по меньшей мере из двух аминокислот, ковалентно связанных амидной связью, независимо от длины и посттрансляционной модификации (например, гликозилирование или фосфорилирование).
[41] "Аминокислоты" обозначаются в настоящем описании посредством либо их широко известных трехбуквенных обозначений, либо посредством однобуквенных обозначений, рекомендованных Комиссией по биохимической номенклатуре IUPAC-IUB. Нуклеотиды, аналогично, могут обозначаться их общепринятыми однобуквенными кодами.
[42] Сокращения, используемые для генетически кодируемых аминокислот, являются общепринятыми и таковы: аланин (Ala или A), аргинин (Are или R), аспарагин (Asn или N), аспартат (Asp или D), цистеин (Cys или C), глутамат (Glu или E), глутамин (Gln или Q), гистидин (His или H), изолейцин (Ile или I), лейцин (Leu или L), лизин (Lys или K), метионин (Met или M), фенилаланин (Phe или F), пролин (Pro или P), серин (Ser или S), треонин (Thr или T), триптофан (Trp или W), тирозин (Tyr или Y) и валин (Val или V).
[43] Когда используют трехбуквенные сокращенные обозначения, если только конкретно не предшествует "L" или "D" или если не очевидно из контекста, в котором используется сокращенное обозначение, аминокислота может быть либо в L-, либо в D-конфигурации у α-углерода (Cα). Например, в то время как "Ala" обозначает аланин без уточнения конфигурации у α-углерода, "D-Ala" и "L-Ala" обозначают D-аланин и L-аланин, соответственно. Когда используется однобуквенные сокращенные обозначения, прописные буквы обозначают аминокислоты в L-конфигурации у α-углерода и строчные буквы обозначают аминокислоты в D-конфигурации у α-углерода. Например, "A" обозначает L-аланин и "a" обозначает D-аланин. Когда полипептидные последовательности представлены в качестве нити однобуквенных или трехбуквенных сокращенных обозначений (или их смесей), последовательности приведены в направлении от амино (N) к карбокси (C) в соответствии с общими правилами.
[44] Сокращенные обозначения, используемые для генетически кодируемых нуклеозидов, являются общепринятыми и таковы: аденозин (A); гуанозин (G); цитидин (C); тимидин (T) и уридин (U). Если не указано конкретно, сокращенно обозначаемые нуклеозиды могут представлять собой либо рибонуклеозиды, либо 2'-дезоксирабонуклеозиды. Уточнение в отношении того, являются ли нуклеозиды рибонуклеозидами или 2'-дезоксирибонуклеозидами приводится индивидуально или в совокупности. Когда последовательности нуклеиновых кислот приведены в качестве нити из однобуквенных сокращенных обозначений, последовательности приводятся в направлении от 5' к 3' в соответствии с общими правилами, и фосфаты не указываются.
[45] Термины "сконструированный", "рекомбинантный", "не встречающийся в природе" и "вариантный", когда их используют в отношении клетки, полинуклеотида или полипептида, относится к материалу, соответствующему природной или нативной форме материала, модифицированному так, что он в ином случае не встречался бы в природе, или идентичному ему, но полученному или происходящему из синтетических материалов и/или путем манипуляции с использованием рекомбинантных способов.
[46] Как используют в рамках изобретения, "дикий тип" и "встречающийся в природе" относятся к форме, встречающейся в природе. Например, последовательность полипептида или полинуклеотида представляет собой последовательность, присутствующую в организме, которая может быть выделена из источника в природе и которая преднамеренно модифицирована путем манипулирования человеком.
[47] "Кодирующая последовательность" относится к той части нуклеиновой кислоты (например, гена), которая кодирует аминокислотную последовательность белка.
[48] Термин "процентная (%) идентичность последовательностей" используют в настоящем описании для обозначения сравнений полинуклеотидов и полипептидов, и ее определяют путем сравнения двух оптимально выровненных последовательностей на протяжении окна сравнения, где часть последовательности полинуклеотида или полипептида в окне сравнения может включать вставки или делеции (т.е. пропуски) по сравнению с эталонной последовательностью для оптимального выравнивания двух последовательностей. Процент можно вычислять путем определения числа положений, в которых идентичные основания нуклеиновых кислот или аминокислотные остатки встречаются в обеих последовательностях с получением количества совпавших положений, деления количества совпавших положений на общее число положений в окне сравнения и умножения результата на 100 с получением процентной идентичности последовательностей. Альтернативно процент можно вычислять путем определения числа положений, в которых либо идентичные основания нуклеиновых кислот или аминокислотные остатки встречаются в обеих последовательностях, либо основания нуклеиновых кислот или аминокислотные остатки выравниваются с использованием пропуска с получением количества совпавших положений, деления количества совпавших положений на общее количество положений в окне сравнения и умножения результата на 100 с получением процента идентичности последовательностей. Специалистам в данной области будет понятно, что существует множество общепризнанных алгоритмов, доступных для выравнивания двух последовательностей. Оптимальное выравнивание последовательностей для сравнения можно проводить, например, с использованием алгоритма локальной гомологии Smith и Waterman (Smith and Waterman, Adv. Appl. Math., 2:482 [1981]), с использование алгоритма выравнивания гомологии Needleman и Wunsch (Needleman and Wunsch, J. Mol. Biol., 48:443 [1970), с использованием способа поиска сходства Pearson и Lipman (Pearson and Lipman, Proc. Natl. Acad. Sci. USA 85:2444 [1988]), с использованием компьютеризованных реализаций этих алгоритмов (например, GAP, BESTFIT, FASTA и TFASTA в GCG Wisconsin Software Package) или посредством визуального исследования, как известно в данной области. Примеры алгоритмов, которые пригодны для определения процентной идентичности последовательностей и сходства последовательностей, включают, но не ограничиваются ими, алгоритмы BLAST и BLAST 2.0, которые описаны Altschul et al. (см. Altschul et al., J. Mol. Biol., 215: 403-410 [1990] и Altschul et al., Nucleic Acids Res., 25:3389-3402 [1977], соответственно). Программное обеспечение для проведения анализов BLAST является общедоступным через web-сайт Национального центра по биотехнологической информации. Этот алгоритм вовлекает идентификацию сначала пар последовательностей с высокой оценкой (HSP) путем идентификации коротких слов длиной W в последовательности запроса, которые либо совпадают, либо удовлетворяют некоторому пороговому показателю T при выравнивании со словом той же длины в последовательности базы данных. T обозначает пороговое значение для соседнего слова (см., Altschul et al., выше). Эти первоначальные наилучшие совпадения для соседних слов выступают в качестве основ для инициации поиска более длинных HSP, содержащих их. Затем наилучшие совпадения удлиняют в обоих направлениях вдоль каждой последовательности насколько, насколько может быть увеличен совокупный показатель выравнивания. Совокупные показатели вычисляют с использованием, для нуклеотидных последовательностей, параметров M (показатель вознаграждения за пару совпавших остатков всегда >0) и N (показатель штрафа за не совпавшие остатки; всегда <0). Для аминокислотных последовательностей для вычисления совокупного показателя используют оценочную матрицу. Удлинение слов с наилучшим совпадением останавливается, когда: совокупный показатель выравнивания снижается на величину X от его максимальной достигнутой величины; совокупный показатель достигает нуля или ниже, вследствие накопления одного или более выравниваний остатков с отрицательной оценкой; или достигается конец любой из последовательностей. Параметры алгоритма BLAST W, T и X определяют чувствительность и скорость выравнивания. Программа BLASTN (для нуклеотидных последовательностей) использует в качестве параметров по умолчанию длину слова (W) 11, ожидание (E) 10, M=5, N=-4, и сравнение обеих цепей. Для аминокислотных последовательностей программа BLASTP использует в качестве параметров по умолчанию длину слова (W) 3, ожидание (E) 10 и оценочную матрицу BLOSUM62 (см., Henikoff и Henikoff, Proc. Natl. Acad. Sci. USA 89:10915 [1989]). Для иллюстративного проведения выравнивания последовательностей и определение % идентичности последовательностей можно использовать программы BESTFIT или GAP в пакете программ GCG Wisconsin Software (Accelrys, Madison WI) с использованием предоставленных параметров по умолчанию.
[49] "Эталонная последовательность" относится к определенной последовательности, используемой в качестве основы для сравнения последовательностей. Эталонная последовательность может представлять собой разновидность более крупной последовательности, например, сегмент полноразмерного гена или полипептидную последовательность. Как правило, эталонная последовательность имеет длину по меньшей мере 20 нуклеотидных или аминокислотных остатков, по меньшей мере 25 остатков, по меньшей мере 50 остатков, по меньшей мере 100 остатков или имеет полную длину нуклеиновой кислоты или полипептида. Поскольку каждый из двух полинуклеотидов или полипептидов может (1) содержать последовательность (т.е. часть полной последовательности), которая является сходной между двумя последовательности, и (2) может дополнительно содержать последовательность, которая различается между двумя последовательности, сравнение последовательностей двух (или более) полинуклеотидов или полипептидов обычно проводят путем сравнения последовательностей двух полинуклеотидов или полипептидов на протяжении "окна сравнения" для идентификации и сравнения локальных областей сходства последовательностей. В некоторых вариантах осуществления "эталонная последовательность" может быть основана на первичной аминокислотной последовательности, где эталонная последовательность представляет собой последовательность, которая может иметь одно или более изменений в первичной последовательности.
[50] "Окно сравнения" относится к схематичному сегменту из по меньшей мере приблизительно 20 последовательных нуклеотидных положений или аминокислотных остатков, где последовательность может быть сравнена с эталонной последовательностью из по меньшей мере 20 последовательных нуклеотидов или аминокислот и где часть последовательности в окне сравнения может включать вставки или делеции (т.е. пропуски), составляющие 20 процентов или менее по сравнению с эталонной последовательностью (которая не включает вставок или делеций) для оптимального выравнивания двух последовательностей. Окно сравнения может составлять более 20 последовательно расположенных остатков и включает, необязательно 30, 40, 50, 100 или более длинные окна.
[51] "Согласно с", "по сравнению с" или "относительно" при использовании в контексте нумерации данной аминокислотной или полинуклеотидной последовательности относится к нумерации остатков определенной эталонной последовательности, когда данную аминокислотную или полинуклеотидную последовательность сравнивают с эталонной последовательностью. Иными словами, номер остатка или положение остатка данного полимера обозначают относительно эталонной последовательности, а не фактического числового положения остатка в данной аминокислотной или полинуклеотидной последовательности. Например, данную аминокислотную последовательность, такую как аминокислотная последовательность сконструированной РНК-полимеразы T7, можно выравнивать с эталонной последовательностью путем внесения пропусков для оптимизации соответствий остатков между двумя последовательностями. В этих случаях, хотя присутствуют пропуски, нумерацию остатка в данной аминокислотной или полинуклеотидной последовательности проводят относительно эталонной последовательности, с которой ее выравнивают.
[52] "Различие аминокислот" или "различие остатков" относится к отличию аминокислотных остатков в положении полипептидной последовательности относительно аминокислотных остатков в соответствующих положениях в эталонной последовательности. Положения различий аминокислот обычно обозначают в настоящем описании как "Xn", где n относится к соответствующему положению в эталонной последовательности, на котором основано различие остатков. Например, "различие остатков в положении X93 по сравнению с SEQ ID NO: 2" относится к различию аминокислотных остатков в положении полипептида, соответствующем положению 93 SEQ ID NO: 2. Таким образом, если эталонный полипептид SEQ ID NO: 2 имеет серин в положении 93, тогда "отличие остатка в положении X93 по сравнению с SEQ ID NO: 2" представляет собой аминокислотную замену в любом остатке, отличном от серина, в положении полипептида, соответствующем положению 93 SEQ ID NO: 2. В большинстве случаев, описанных в настоящем описании, конкретное различие аминокислотных остатков в данном положении обозначается как "XnY", где "Xn" обозначает соответствующее положение, как описано выше, и "Y" представляет собой однобуквенный идентификатор аминокислоты, находящейся в сконструированном полипептиде (т.е. остаток, отличный от эталонного полипептида). В некоторых случаях (например, в таблицах, приведенных в примерах настоящего описания), настоящее изобретение также относится к конкретным аминокислотным различиям, обозначаемым посредством общепринятого обозначения "AnB", где A представляет собой однобуквенный идентификатор остатка в эталонной последовательности, "n" представляет собой количество положений остатков в эталонной последовательности и B представляет собой однобуквенный идентификатор замены остатков в последовательности сконструированного полипептида. В некоторых случаях полипептид по настоящему изобретению может включать одно или более отличий аминокислотных остатков относительно эталонной последовательности, которые указываются посредством перечня указанных положений, где присутствуют различия остатков относительно эталонной последовательности. В некоторых вариантах осуществления, когда более одной аминокислоты может использоваться в конкретном положении остатка полипептида, различные аминокислотные остатки, которые могут использоваться, разделены "/" (например, X10H/X10P или X10H/P). В некоторых вариантах осуществления варианты ферментов включают более одной замены. Эти замены разделены слешем для простоты прочтения (например, C14A/K122A). Настоящая заявка включает сконструированные полипептидные последовательности, содержащие одно или более аминокислотных различий, которые включают либо консервативные, либо неконсервативные аминокислотные замены, или и те, и другие.
[53] "Консервативная аминокислотная замена" относится к замене остатка отличающимся остатком, имеющим сходную боковую цепь и, таким образом, она обычно вовлекает замену аминокислоты в полипептиде аминокислотами в пределах одного и того же или сходного определенного класса аминокислот. В качестве неограничивающего примера, аминокислота с алифатической боковой цепью может быть заменена другой алифатической аминокислотой (например, аланин, валин, лейцин и изолейцин); аминокислота с гидроксильной боковой цепью заменена другой аминокислотой с гидрофильной боковой цепью (например, серином и треонином); аминокислоты, имеющие ароматические боковые цепи, заменены другой аминокислотой, имеющей ароматическую боковую цепь (например, фенилаланин, тирозин, триптофан и гистидин); аминокислота с основной боковой цепью заменена другой аминокислотой с основной боковой цепью (например, лизин и аргинин); аминокислота с кислотной боковой цепью заменена другой аминокислотой с кислотной боковой цепью (например, аспарагиновая кислота или глутаминовая кислота); и/или гидрофобная или гидрофильная аминокислота заменена другой гидрофобной или гидрофильной аминокислотой, соответственно.
[54] "Неконсервативная замена" относится к замене аминокислоты в пептиде аминокислотой со значительно отличающимися свойствами боковой цепи. В случае неконсервативных замен могут использоваться аминокислоты между, а не в пределах, определенными группами, и они влияют на (a) структуру пептидного остова в области замещения (например, пролин на глицин) (b) заряд или гидрофобность или (c) объем боковой цепи. В качестве неограничивающего примера иллюстративная неконсервативная замена может представлять собой кислотную аминокислоту, замещенную основной или алифатической аминокислотой; ароматическую аминокислоту, замещенную небольшой аминокислотой; и гидрофобную аминокислоту, замещенную гидрофобной аминокислотой.
[55] "Делеция" относится к модификации полипептида путем удаления одной или более аминокислот из эталонного полипептида. Делеции могут включать удаление на 1 или более аминокислот, 2 или более аминокислот, 5 или более аминокислот, 10 или более аминокислот, 15 или более аминокислоты, или 20 или более аминокислот, вплоть до 10% от общего количества аминокислот, или вплоть до 20% от общего количества аминокислот, составляющих эталонный фермент, при сохранении ферментативной активности и/или сохранении усовершенствованных свойств сконструированного фермента. Делеции могут быть направлены на внутренние части и/или концевые части полипептида. В различных вариантах осуществления делеция может включать непрерывный сегмент или может прерываться.
[56] "Инсерция" относится к модификации полипептида путем присоединения одной или более аминокислот из эталонного полипептида. Инсерции могут быть проведены во внутренних частях полипептида или на C-конце или N-конце. Инсерции, как используют в рамках изобретения, включают слитые белки, как известно в данной области. Инсерция может представлять собой непрерывный сегмент из аминокислот или может быть разделена одной или более аминокислотами встречающегося в природе полипептида.
[57] "Выделенный полипептид" относится к полипептиду, который по существу отделен от других компонентов, которые обычно сопровождают его (например, белок, липиды и полинуклеотиды). Термин охватывает полипептиды, которые извлечены или очищены из их встречающегося в природе окружения или экспрессирующей системы (например, клетка-хозяин или синтез in vitro). Рекомбинантные полипептиды РНК-полимеразы T7 могут присутствовать в клетке, могут присутствовать в клеточной среде или могут быть получены в различных формах, таких как лизаты или выделенные препараты. По существу, в некоторых вариантах осуществления рекомбинантные полипептиды РНК-полимеразы T7 могут представлять собой выделенные полипептиды.
[58] "По существу чистый полипептид" относится к композиции, в которой полипептидный компонент представляет собой преобладающий присутствующий компонент (т.е. на молярной или весовой основе он присутствует в большем количестве, чем любой другой отдельный макромолекулярный компонент в композиции), и обычно представляет собой по существу очищенную композицию, когда данный компонент составляет по меньшей мере приблизительно 50 процентов макромолекулярных компонентов на молярной основе или в % по массе. Как правило, по существу чистая композиция РНК-полимеразы T7 содержит 60% или более, приблизительно 70% или более, приблизительно 80% или более, приблизительно 90% или более, приблизительно 95% или более, и приблизительно 98% или всех макромолекулярных компонентов на молярной основе или в % по массе, присутствующих в композиции. В некоторых вариантах осуществления рассматриваемый компонент очищают по существу до однородности (т.е. загрязняющие компоненты не могут быть обнаружены общепринятыми способами детекции), где композиция по существу состоит из одного макромолекулярного компонента. Компоненты растворителей, низкомолекулярные соединения (<500 Дальтон) и компоненты в виде элементарных ионов не считаются макромолекулярными компонентами. В некоторых вариантах осуществления выделенные рекомбинантные полипептиды РНК-полимеразы T7 представляют собой по существу чистые полипептидные композиции.
[59] "Усовершенствованное свойство фермента" РНК-полимеразы T7 относится к сконструированному полипептиду РНК-полимеразы T7, который демонстрирует улучшение какого-либо ферментного свойства по сравнению с эталонным полипептидом РНК-полимеразы T7 и/или полипептидом РНК-полимеразы T7 дикого типа и/или другим сконструированным полипептидом РНК-полимеразы T7. Усовершенствованные свойства включают, но не ограничиваются ими, такие свойства, как увеличенная селективность в отношении аналога кэпа относительно GTP, увеличенная точность репликации, увеличенный выход РНК, увеличенная экспрессия белка, увеличенная термоактивность, увеличенная термостабильность, увеличенная зависимая от pH активность, увеличенная стабильность, увеличенная ферментативная активность, увеличенная субстратная специфичность или аффинность, увеличенная удельная активность, увеличенная резистентность к субстрату или ингибирование конечного продукта (включая пирофосфат), увеличенная химическая стабильность, увеличенная стабильность в растворителях, увеличенная толерантность к кислотным или основным значениям pH, увеличенная толерантность к протеолитической активности (т.е. сниженная чувствительность к протеолизу), сниженная агрегация, увеличенная растворимость и измененный профиль температур.
[60] "Увеличенная ферментативная активность" или "усиленная каталитическая активность" относится к улучшенному свойству сконструированной РНК-полимеразы T7, которое может отражаться увеличением удельной активности (например, продуцированный продукт/время/масса белка) или увеличением процентного конвертирования субстрата в продукт (например, процентное конвертирование исходного количества субстрата в продут за определенный период времени с использованием определенного количества варианта РНК-полимеразы T7 по сравнению с эталонной РНК-полимеразой T7. Иллюстративные способы определения ферментативной активности приведены в примерах. Может изменяться любое свойство, касающееся ферментативной активности.
[61] "Жесткость гибридизации" относится к условиям гибридизации, таким как условия промывания, при гибридизации нуклеиновых кислот. Как правило, реакции гибридизации проводят в условиях более низкой жесткости, после чего проводят промывания варьирующей, но более высокой жесткости. Термин "умеренно жесткая гибридизация" относится к условиям, которые позволяют ДНК-мишени связать комплементарную нуклеиновую кислоту, которая обладает приблизительно 60% идентичностью, предпочтительно приблизительно 75% идентичностью, приблизительно 85% идентичностью с ДНК-мишенью, с более чем приблизительно 90% идентичностью с полинуклеотидом-мишенью. Иллюстративные условия умеренной жесткости представляют собой условия, эквивалентные гибридизации в 50% формамиде, 5× растворе Денхарта, 5×SSPE, 0,2% SDS при 42°C, с последующим промыванием в 0,2×SSPE, 0,2% SDS, при 42°C. "Гибридизация высокой жесткости" относится, главным образом, к условиям, которые приблизительно на 10°C или менее ниже температуры плавления Tm при определении в условиях раствора для определенной полинуклеотидной последовательности. В некоторых вариантах осуществления условия высокой жесткости относятся к условиям, которые позволяют гибридизацию только тех последовательностей нуклеиновых кислот, которые образуют стабильные гибриды в 0,018 M NaCl при 65°C (т.е. если гибрид не является стабильным в 0,018 M NaCl при 65°C, предусматривается, что он не будет стабильным в условиях высокой жесткости). Условия высокой жесткости могут быть обеспечены, например, гибридизацией в условиях, эквивалентных 50% формамиду, 5× раствору Денхарта, 5×SSPE, 0,2% SDS при 42°C, с последующим промыванием в 0,1×SSPE и 0,1% SDS при 65°C. Другими условиями высокой жесткости является гибридизация в условиях, эквивалентных гибридизации в 5X SSC, содержащем 0,1% (масс.:об.) SDS, при 65°C, и промывание в 0,1x SSC, содержащем 0,1% SDS, при 65°C. Другие условиях гибридизации высокой жесткости, а также условия умеренной жесткости, описаны в ссылках, цитированных выше.
[62] "Кодон-оптимизированный" относится к изменениям в кодонах полинуклеотида, кодирующего белок, на кодоны, предпочтительно используемые в конкретном организме, так что кодируемый белок более эффективно экспрессируется в представляющем интерес организме. Хотя генетический код является вырожденным ввиду того, что большинству аминокислот соответствует несколько кодонов, называемых "синонимами" или "синонимическими" кодонами, хорошо известно, что использование кодонов конкретными организмами является неслучайным и предпочтительным с точки зрения конкретных кодоновых триплетов. Это предпочтение использования кодонов может быть более высоким для данного гена, генов с общей функцией или происхождением, высоко экспрессируемых белков против белков с низким числом копий, и составные области, кодирующие белки, генома организма. В некоторых вариантах осуществления полинуклеотиды, кодирующие ферменты РНК-полимеразы T7, могут быть кодон-оптимизированными для оптимального продуцирования из организма-хозяина, выбранного для экспрессии.
[63] "Последовательность контроля" относится в настоящем описании ко всем компонентам, которые являются необходимыми или преимущественными для экспрессии полинуклеотида и/или полипептида, описанного в настоящей заявке. Каждая последовательность контроля может быть нативной или чужеродной для последовательности нуклеиновой кислоты, кодирующей полипептид. Такие последовательности контроля включают, но не ограничиваются ими, лидерную последовательность, последовательность полиаденилирования, последовательность пропептида, промоторную последовательность, последовательность сигнального пептида, последовательность инициации и терминатор транскрипции. Как минимум, последовательности контроля включают промотор и стоп-сигналы транскрипции и трансляции. Последовательности контроля могут быть предоставлены с линкерами для внесения специальных участков рестрикции, облегчающих лигирование последовательностей контроля с кодирующей областью последовательности нуклеиновой кислоты, кодирующей полипептид.
[64] "Функционально связанный" определяют в настоящем описании как конфигурацию, в которой последовательность контроля соответствующим образом помещена (т.е. в функциональной взаимосвязи) в таком положении относительно представляющего интерес полинуклеотида, что последовательность контроля направляет или регулирует экспрессию представляющего интерес полинуклеотида и/или полипептида.
[65] "Промоторная последовательность" относится к последовательности нуклеиновой кислоты, которая распознается клеткой-хозяином для экспрессии представляющего интерес полинуклеотида, такого как кодирующая последовательность. Промоторная последовательность содержит последовательности контроля транскрипции, которые опосредуют экспрессию представляющего интерес полинуклеотида. Промотор может представлять собой любую последовательность нуклеиновой кислоты, которая демонстрирует транскрипционную активность в выбранной клетке-хозяине, включая мутантный, укороченный и гибридный промотор, и может быть получена из генов, кодирующих внеклеточные или внутриклеточные полипептиды, либо гомологичные, либо гетерологичные для клетки-хозяина.
[66] "Подходящие условия реакции" относятся к условиям в реакционном растворе для ферментативного превращения (например, диапазоны загрузки фермента, загрузки субстрата, температура, pH, буферы, сорастворители и т.д.), в которых полипептид РНК-полимеразы T7, описанный в настоящей заявке, способен конвертировать субстрат в требуемое соединение-продукт.
[67] "Субстрат" в контексте процесса ферментативной реакции конвертирования относится к соединению или молекуле, на которую действует полипептид РНК-полимеразы T7.
[68] "Продукт" в контексте процесса ферментативного превращения относится к соединению или молекуле, образующимся в результате действия полипептида РНК-полимеразы T7 на субстрат.
[69] Как используют в рамках изобретения термин "культивирование" относится к выращиванию популяции микробных клеток в любых подходящих условиях (например, с использованием жидкости, геля или твердой среды).
[70] Рекомбинантные полипептиды можно получать любыми подходящими способами, известными в данной области. Гены, кодирующие представляющий интерес полипептид дикого типа, можно клонировать в векторы, такие как плазмиды, и экспрессировать в желаемых хозяевах, таких как E. coli, S. Cerevisiae и т.д. Варианты рекомбинантных полипептидов можно получать различными способами, известными в данной области. Действительно, существует широкое множество различных способов мутагенеза, хорошо известных специалистам в данной области. Кроме того, также доступны наборы для мутагенеза от множества коммерческих молекулярно-биологических поставщиков. Доступны способы для проведения конкретных замен в определенных аминокислотах (сайт-направленных), конкретных или случайных мутаций в локализованной области гена (региоспецифических) или случайного мутагенеза по всему гена (например, насыщающий мутагенез). Специалистам в данной области хорошо известны многочисленные подходящие способы получения вариантов ферментов, включая, но не ограничиваясь ими, сайт-направленный мутагенез одноцепочечной ДНК или двухцепочечной ДНК с использованием ПЦР, кассетного мутагенеза, синтеза генов, ПЦР с пониженной точностью, шаффлинга и химического насыщающего мутагенеза, или любого другого подходящего способа, известного в данной области. Неограничивающие примеры способов, используемых для инженерии ДНК и белков, приведены в следующих патентах: патент США №6117679; патент США №6420175; патент США №6376246; патент США №6586182; патент США №7747391; патент США №7747393; патент США №7783428 и патент США №8383346. После получения вариантов их можно подвергать скринингу в отношении любого желаемого свойства (например, высокая или увеличенная активность, или низкая или сниженная активность, увеличенная термическая активность, увеличенная термическая стабильность и/или стабильность при кислых значениях pH и т.д.).
[71] В некоторых вариантах осуществления пригодными являются "рекомбинантные полипептиды РНК-полимеразы T7" (также обозначаемые в настоящем описании как "сконструированные полипептиды РНК-полимеразы T7", "варианты ферментов РНК-полимераз T7" и "варианты РНК-полимераз T7").
[72] Как используют в рамках изобретения, "вектор" представляет собой конструкцию ДНК для введения последовательности ДНК в клетку. В некоторых вариантах осуществления вектор представляет собой экспрессирующий вектор, который функционально связан с подходящей последовательностью контроля, способной обеспечивать экспрессию в подходящем хозяине полипептида, кодируемого в последовательности ДНК. В некоторых вариантах осуществления "экспрессирующий вектор" имеет промоторную последовательность, функционально связанную с последовательностью ДНК (например, трансгеном) для запуска экспрессии в клетке-хозяине и в некоторых вариантах осуществления также содержит последовательность терминатора транскрипции.
[73] Как используют в рамках изобретения, термин "экспрессия" включает любую стадию, вовлеченную в продуцирование полипептида, включая, но не ограничиваясь ими, транскрипцию, посттранскрипционную модификацию, трансляцию и посттрансляционную модификацию. В некоторых вариантах осуществления термин также охватывает секрецию полипептида из клетки.
[74] Как используют в рамках изобретения, термин "продуцирует" относится к продуцированию белков и/или других соединений клетками. Подразумевается, что термин охватывает любую стадию, вовлеченную в продуцирование полипептидов, включая, но не ограничиваясь ими, транскрипцию, посттранскрипционную модификацию, трансляцию и посттрансляционную модификацию. В некоторых вариантах осуществления термин также охватывает секрецию полипептида из клетки.
[75] Как используют в рамках изобретения, аминокислотная или нуклеотидная последовательность (например, промоторная последовательность, сигнальный пептид, терминаторная последовательность и т.д.) является "гетерологичной" для другой последовательности, с которой она функционально связана, если эти две последовательности не ассоциированы в природе.
[76] Как используют в рамках изобретения, термины "клетка-хозяин" и "штамм-хозяин" относятся к подходящим хозяевам для экспрессирующих векторов, содержащих ДНК, описанную в настоящем описании (например, полинуклеотиды, кодирующие варианты РНК-полимеразы T7). В некоторых вариантах осуществления клетки-хозяева представляют собой прокариотические или эукариотические клетки, трансформированные или трансфицированные векторами, сконструированными с использованием способов рекомбинантных ДНК, известных в данной области.
[77] Термин "аналог", когда его используют в отношении полипептида, означает полипептид, обладающий более чем 70% идентичностью последовательности, но менее чем 100% идентичностью последовательности (например, более чем 75%, 78%, 80%, 83%, 85%, 88%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% идентичностью последовательности) с эталонным полипептидом. В некоторых вариантах осуществления аналог означает полипептиды, которые содержат один или более не встречающихся в природе аминокислотных остатков, включая, но не ограничиваясь ими, гомоаргинин, орнитин и норвалин, а также встречающиеся в природе аминокислоты. В некоторых вариантах осуществления аналог также включает один или более остатков D-аминокислот и непептидных связей между двумя или более аминокислотными остатками.
[78] Термин "эффективное количество" означает количество, достаточное для достижения желаемого результата. Специалист в данной области может определить эффективное количество с использованием стандартного экспериментирования.
[79] Термины "выделенный" и "очищенный" используют для обозначения молекулы (например, выделенная нуклеиновая кислота, полипептид и т.д.) или другого компонента, который отделен от по меньшей мере одного другого компонента, с которым он ассоциирован в природе. Термин "очищенный" не требует абсолютной чистоты, скорее он предполагает относительное определение.
[80] Как используют в рамках изобретения, "композиция" и "состав" охватывают продукты, содержащие по меньшей мере одну сконструированную РНК-полимеразу T7 по настоящему изобретению, предназначенную для любого подходящего применения (например, научная деятельность, диагностика и т.д.).
[81] Термин "транскрипция" используют для обозначения процесса, в котором часть ДНК-матрицы копируется в РНК под действием фермента РНК-полимеразы.
[82] Термин "ДНК-матрица" используют для обозначения двухцепочечной или одноцепочечной молекулы ДНК, включающей промоторную последовательность и последовательность, кодирующую РНК-продукт транскрипции.
[83] Термин "промотор" используют для обозначения последовательности ДНК, которая распознается РНК-полимеразой в качестве участка начала транскрипции. Промотор привлекает РНК-полимеразу и в случае РНК-полимеразы T7 определяет участок начала транскрипции.
[84] Термин "РНК-полимераза" используют для обозначения ДНК-направленной РНК-полимеразы, которая копирует ДНК-матрицу в РНК-полинуклеотид, путем пошагового включения нуклеотидтрифосфатов в растущий полимер РНК.
[85] Термины "матричная РНК" и "мРНК" используют для обозначения молекул РНК, которые кодируют белок. Этот белок декодируется в ходе трансляции.
[86] Термины "7-метилгуанозиновый кэп", "7meG", "кэп five-prime" и "5'-кэп" используют в отношении определенной модифицированной нуклеотидной структуры, присутствующей на 5'-конце эукариотических мРНК. Структура 7-метилгуанозинового кэпа связывается через 5'-5'-трифосфатную связь с первым нуклеотидом в мРНК. In vivo эта структура кэпа присоединяется к 5'-концу образующейся мРНК посредством последовательной активности множества ферментов. In vitro кэп может быть включен прямо при начале транскрипции РНК-полимеразой с использованием аналога кэпа.
[87] Термин "аналог кэпа" относится к динуклеотиду, содержащему 5'-5' ди-, три- или тетра-фосфатную связь. Один конец динуклеотида терминируется либо остатком гуанозина, либо остатком замещенного гуанозина; именно с этой формы конца РНК-полимераза будет инициировать транскрипцию путем удлинения от 3'-гидроксила. Другой конец динуклеотида представляет собой гуанозин, который имитирует структуру эукариотического кэпа и, как правило, имеет замещение 7-метилом, 7-бензилом или 7-этилом и/или замещение 7-аминометилом или 7-аминоэтилом. В некоторых случаях этот нуклеотид также является метоксизамещенным на 3'-гидроксильной группе для предупреждения инициации транскрипции с конца кэпа молекулы.
[88] Термины "ARCA" и "антиреверсный аналог кэпа" относятся к химически модифицированным формам аналогов кэпа, предназначенным для максимизации эффективной трансляции in vitro путем обеспечения надлежащего включения аналога кэпа в транскрипт в правильной ориентации. Эти аналоги применимы для усиления трансляции. В некоторых вариантах осуществления применимы ARCA, известные в данной области (например, Peng et al., Org. Lett., 4:161-164 [2002]).
[89] Термин "рибопереключатель" используют для обозначения аутокаталитического РНК-фермента, который расщепляет сам себя или другую РНК в присутствии лиганда.
[90] Термин "точность" используют для обозначения безошибочности РНК-полимеразы при транскрибировании или копировании ДНК-матрицы в РНК-полинуклеотид. Неточная транскрипция может приводить к однонуклеотидным полиморфизмам (SNP) или инсерциям-делециям.
[91] Термины "однонуклеотидный полиморфизм" или "SNP" относятся к изменению нуклеотида, присутствующего в единичном положении полинуклеотида. В контексте транскрипции SNP могут быть результатом неправильного включения некомплементарного рибонуклеотида (A, C, G или U) РНК-полимеразой в некотором положении на ДНК-матрице.
[92] Термин "инсерция-делеция" используют для обозначения инсерции или делеции одного или более полинуклеотидов. В контексте транскрипции РНК-полимеразой ошибки инсерций-делеций могут возникать в результате присоединения одного или более дополнительных рибонуклеотидов или не включения одного или более нуклеотидов в некотором положении на ДНК-матрице.
[93] Термин "селективность" используют для обозначения признака наличия у фермента более высокой активности против одного субстрата по сравнению с другим субстратом в ходе катализируемой реакции. В контексте котранскрипционного кэпирования РНК-полимераза может иметь высокую или низкую селективность в отношении аналога кэпа относительно GTP.
[94] Термин "неорганическая пирофосфатаза" используют для обозначения фермента, который деградирует неорганический пирофосфат в ортофосфат.
Активность сконструированной РНК-полимеразы T7:
[95] В некоторых вариантах осуществления сконструированная РНК-полимераза T7, которая демонстрирует усовершенствованное свойство, обладает по меньшей мере приблизительно 85%, по меньшей мере приблизительно 88%, по меньшей мере приблизительно 90%, по меньшей мере приблизительно 91%, по меньшей мере приблизительно 92%, по меньшей мере приблизительно 93%, по меньшей мере приблизительно 94%, по меньшей мере приблизительно 95%, по меньшей мере приблизительно 96%, по меньшей мере приблизительно 97%, по меньшей мере приблизительно 98%, по меньшей мере приблизительно 99% или приблизительно 100% идентичностью аминокислотной последовательности с SEQ ID NO: 4 и/или 15, и различием аминокислотных остатков по сравнению с SEQ ID NO: 4 и/или 15, в одном или более положениях аминокислот (как например, в 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 14, 15, 20 или более положениях аминокислот по сравнению с SEQ ID NO: 4 и/или 15, или последовательностью, обладающей по меньшей мере 85%, по меньшей мере 88%, по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или более идентичностью аминокислотной последовательности с SEQ ID NO: 4 и/или 15). В некоторых вариантах осуществления различие остатков по сравнению с SEQ ID NO: 4 в одном или более положениях включает по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более консервативных аминокислотных замен. В некоторых вариантах осуществления сконструированный полипептид РНК-полимеразы T7 представляет собой полипептид, приведенный в таблице 5.3, 5.5 и/или 5.6. В некоторых вариантах осуществления сконструированный полипептид РНК-полимеразы T7 выбран из SEQ ID NO: 4, 15, 17, 19, 21, 23, 25, 27, 29, 32, 33, 35, 37 и/или 39. В некоторых вариантах осуществления различие остатков по сравнению с SEQ ID NO: 15 в одном или более положениях может включать по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более консервативных аминокислотных замен. В некоторых вариантах осуществления сконструированный полипептид РНК-полимеразы T7 представляет собой полипептид, приведенный в таблице 5.4. В некоторых вариантах осуществления сконструированный полипептид РНК-полимеразы T7 выбран из SEQ ID NO: 4, 15, 17, 19, 21, 23, 25, 27, 29, 32, 33, 35, 37 и/или 39.
[96] В некоторых вариантах осуществления сконструированный полипептид РНК-полимеразы T7 включает функциональный фрагмент сконструированного полипептида РНК-полимеразы T7, охватываемый изобретением. Функциональные фрагменты обладают по меньшей мере 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% активности сконструированного полипептида РНК-полимеразы T7, из которого они происходят (т.е. исходной сконструированной РНК-полимеразы T7). Функциональный фрагмент содержит по меньшей мере 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% и даже 99% исходной последовательности сконструированной РНК-полимеразы T7. В некоторых вариантах осуществления функциональный фрагмент укорочен менее чем на 5, менее чем на 10, менее чем на 15, менее чем на 10, менее чем на 25, менее чем на 30, менее чем на 35, менее чем на 40, менее чем на 45 и менее чем на 50 аминокислот.
Полинуклеотиды, кодирующие сконструированные полипептиды, экспрессирующие векторы и клетки-хозяева:
[97] Настоящее изобретение относится к полинуклеотидам, кодирующим сконструированные полипептиды РНК-полимеразы T7, описанные в настоящем описании. В некоторых вариантах осуществления полинуклеотиды функционально связаны с одной или более гетерологичными регуляторными последовательностями, которые контролируют экспрессию генов, с образованием рекомбинантного полинуклеотида, способного экспрессировать полипептид. Экспрессирующие конструкции, содержащие гетерологичный полинуклеотид, кодирующий сконструированные полипептиды РНК-полимеразы T7, можно вводить в соответствующие клетки-хозяева для экспрессии соответствующего полипептида РНК-полимеразы T7.
[98] Как будет понятно специалисту в данной области, доступность белковой последовательности и знание кодонов, соответствующих различным аминокислотам, обеспечивают описание всех полинуклеотидов, способных кодировать рассматриваемые полипептиды. Вырожденность генетического когда, где одни и те же аминокислоты кодируются альтернативными или синонимическими кодонами, позволяет получение чрезвычайно большого количества нуклеиновых кислот, все из которых кодируют сконструированный полипептид РНК-полимеразы T7. Таким образом, зная конкретную аминокислотную последовательность, специалисты в данной области могут получить любое количество различных нуклеиновых кислот, просто модифицируя последовательность одного или более кодонов так, чтобы аминокислотная последовательность белка не изменялась. В этом отношении настоящее изобретение конкретно предусматривает каждый возможный вариант полинуклеотидов, который может быть сконструирован так, чтобы он кодировал полипептиды, описанные в настоящем описании, посредством выбора комбинаций на основе выбора возможных кодонов, и все такие варианты считаются конкретно раскрытыми для любого полипептида, описанного в настоящем описании, включая варианты, приведенные в таблицах 5.3, 5.4, 5.5 и/или 5.6.
[99] В различных вариантах осуществления кодоны предпочтительно выбирают так, чтобы они были подходящими для клетки-хозяина, в которой будет продуцироваться белок. Например, для экспрессии в бактериях используют предпочтительные кодоны, используемые бактериями. Следовательно, кодон-оптимизированные полинуклеотиды, кодирующие сконструированные полипептиды РНК-полимеразы T7, содержат предпочтительные кодоны в приблизительно 40%, 50%, 60%, 70%, 80% или более 90% положений кодонов полноразмерной кодирующей области.
[100] В некоторых вариантах осуществления, как описано выше, полинуклеотид кодирует сконструированный полипептид, обладающий активностью РНК-полимеразы T7, со свойствами, описанными в настоящем описании, где полипептид содержит аминокислотную последовательность, обладающую по меньшей мере 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичностью с эталонной последовательностью, выбранной из SEQ ID NO: 4 и 15, или аминокислотную последовательность любого варианта, как описано в таблицах 5.3, 5.4., 5.5 и/или 5.6, и одно или более различий остатков по сравнению с эталонным полипептидом SEQ ID NO: 17, 19, 21, 23, 25, 27, 29, 32, 33, 35, 37 и/или 39, или аминокислотную последовательность любого варианта, как описано в таблицах 5.3, 5.4, 5.5 и/или 5.6 (например 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более положений аминокислотных остатков). В некоторых вариантах осуществления эталонная последовательность выбрана из SEQ ID NO: 4 и/или 15. В некоторых вариантах осуществления полинуклеотид кодирует сконструированный полипептид, обладающий активностью РНК-полимеразы T7, со свойствами, описанными в настоящем описании, где полипептид содержит аминокислотную последовательность, обладающую по меньшей мере 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичностью последовательности с эталонной последовательностью SEQ ID NO: 4 и/или 15, и одно или более отличий остатков по сравнению с SEQ ID NO: 4 и/или 15 в положениях остатков, выбранных из положений остатков, приведенных в таблицах 5.3, 5.4, 5.5 и/или 5.6 при оптимальном выравнивании с полипептидом SEQ ID NO: 4 и/или 15.
[101] В некоторых вариантах осуществления полинуклеотид, кодирующий сконструированные полипептиды РНК-полимеразы T7, содержит полинуклеотидную последовательность, выбранную из полинуклеотидной последовательности, кодирующей SEQ ID NO: 4, 15, 17, 19, 21, 23, 25, 27, 29, 32, 33, 35, 37 и 39. В некоторых вариантах осуществления полинуклеотид, кодирующий сконструированный полипептид РНК-полимеразы T7, обладает по меньшей мере 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 93%, 95%, 96%, 97%, 98%, 99% идентичностью нуклеотидных остатков с SEQ ID NO: 3, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36 и 38. В некоторых вариантах осуществления полинуклеотид, кодирующий сконструированные полипептиды РНК-полимеразы T7, содержит полинуклеотидную последовательность, выбранную из SEQ ID NO: 3, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 36 и 38.
[102] В некоторых вариантах осуществления полинуклеотиды способные гибридизоваться в условиях высокой жесткости с эталонной полинуклеотидной последовательностью, выбранной из SEQ ID NO: 3 и/или 14, или комплементарной ей последовательностью, или полинуклеотидной последовательностью, кодирующей любой из вариантов полипептидов РНК-полимеразы T7, описанных в настоящем описании. В некоторых вариантах осуществления полинуклеотид, способный гибридизоваться в условиях высокой жесткости, кодирует полипептид РНК-полимеразы T7, содержащий аминокислотную последовательность, которая обладает одним или более различиями остатков по сравнению с SEQ ID NO: 4 и/или 15 в положениях остатков, выбранных из любых положений, как указано в таблице 5.3, 5.4, 5.5 и/или 5.6.
[103] В некоторых вариантах осуществления выделенный полинуклеотид, кодирующий любой из сконструированных полипептидов РНК-полимеразы T7, описанных в настоящем описании, подвергают различным манипуляциям для обеспечения экспрессии полипептида. В некоторых вариантах осуществления полинуклеотиды, кодирующие полипептиды, предоставляют в качестве экспрессирующих векторов, в которых присутствует одна или более последовательностей контроля для регуляции экспрессии полинуклеотидов и/или полипептидов. Манипулирование выделенным полинуклеотидом перед его встраиванием в вектор может быть желательным или необходимым в зависимости от экспрессирующего вектора. Способы модификации полинуклеотидов и последовательностей нуклеиновых кислот с использованием способов рекомбинантных ДНК хорошо известны в данной области.
[104] В некоторых вариантах осуществления последовательности контроля включают, среди прочих последовательностей, промоторы, лидерные последовательности, последовательности полиаденилирования, последовательности пропептидов, последовательности сигнальных пептидов и терминаторы транскрипции. Как известно в данной области, подходящие промоторы могут быть выбраны, исходя из используемых клеток-хозяев. Иллюстративные промоторы для клеток-хозяев в виде нитчатых грибов включают промоторы, полученные из генов для амилазы TAKA Aspergillus oryzae, аспарагиновой протеазы Rhizomucor miehei, нейтральной альфа-амилазы Aspergillus niger, устойчивой к кислотам альфа-амилазы Aspergillus niger, глюкоамилазы Aspergillus niger или Aspergillus awamori (glaA), липазы Rhizomucor miehei, щелочной протеазы Aspergillus oryzae, триозофосфатизомеразы Aspergillus oryzae, ацетамидазы Aspergillus nidulans и трипсин-подобной протеазы Fusarium oxysporum (см., например, WO 96/00787), а также промотор NA2-tpi (гибрид промоторов из генов нейтральной альфа-амилазы Aspergillus niger и триозофосфатизомеразы Aspergillus oryzae), и их мутантных, укороченных и гибридных промоторов. Иллюстративные промоторы дрожжевых клеток могут представлять собой промоторы из генов энолазы Saccharomyces cerevisiae (ENO-1), галактокиназы Saccharomyces cerevisiae (GAL1), алкогольдегидрогеназы/глицеральдегид-3-фосфатдегидрогеназы Saccharomyces cerevisiae (ADH2/GAP) и 3-фосфоглицераткиназы Saccharomyces cerevisiae. В данной области известны другие пригодные промоторы для дрожжевых клеток (см., например, Romanos et al., Yeast 8:423-488 [1992]). Иллюстративные промоторы для применения в клетках млекопитающих включают, но не ограничиваются ими, промоторы из цитомегаловируса (CMV), вакуолизирующего вируса обезьян 40 (SV40), из фосфоглицераткиназы Homo sapiens, бета-актина, фактора элонгации 1a или глицеральдегид-3-фосфатдегидрогеназы, или из β-актина Gallus gallus.
[105] В некоторых вариантах осуществления последовательность контроля представляет собой подходящую последовательность терминатора транскрипции - последовательность, распознаваемую клеткой-хозяином для терминации транскрипции. Последовательность терминатора функционально связана с 3'-концом последовательности нуклеиновой кислоты, кодирующей полипептид. В рамках настоящего изобретения применим любой терминатор, который является функциональным в клетке-хозяине. Например, иллюстративные терминаторы транскрипции для клеток-хозяев в виде нитчатых грибов могут быть получены из генов амилазы TAKA Aspergillus oryzae, глюкоамилазы Aspergillus niger, антранилатсинтазы Aspergillus nidulans, альфа-глюкозидазы Aspergillus niger и трипсин-подобной протеазы Fusarium oxysporum. Иллюстративные терминаторы для дрожжевых клеток-хозяев могут быть получены из генов энолазы Saccharomyces cerevisiae, цитохрома С Saccharomyces cerevisiae (CYC1) и глицеральдегид-3-фосфатдегидрогеназы Saccharomyces cerevisiae. Другие пригодные терминаторы для дрожжевых клеток-хозяев известны в данной области (см., например, Romanos et al., выше). Иллюстративные терминаторы для клеток млекопитающих включают, но не ограничиваются ими, терминаторы из цитомегаловируса (CMV), вакуолизирующего вируса обезьян 40 (SV40) или из гормона роста Homo sapiens.
[106] В некоторых вариантах осуществления последовательность контроля представляет собой подходящую лидерную последовательность - нетранслируемую область мРНК, которая важна для трансляции клеткой-хозяином. Лидерная последовательность функционально связана с 5'-концом последовательности нуклеиновой кислоты, кодирующей полипептид. Можно использовать любую лидерную последовательность, которая функциональна в выбранной клетке-хозяине. Иллюстративные лидерные последовательности для клеток-хозяев в виде нитчатых грибов получают из генов амилазы TAKA Aspergillus oryzae и триозофосфатизомеразы Aspergillus nidulans. Подходящие лидерные последовательности для дрожжевых клеток-хозяев включают, но не ограничиваются ими, лидерные последовательности, полученные из генов энолазы Saccharomyces cerevisiae (ENO-1), 3-фосфоглицераткиназы Saccharomyces cerevisiae, альфа-фактора Saccharomyces cerevisiae и алкогольдегидрогеназы/глицеральдегид-3-фосфатдегидрогеназа Saccharomyces cerevisiae (ADH2/GAP).
[107] Последовательность контроля также может представлять собой последовательность полиаденилирования - последовательность, функционально связанную с 3'-концом последовательности нуклеиновой кислоты и которая при транскрипции распознается клеткой-хозяином в качестве сигнала для присоединения остатков полиаденозина к транскрибируемой мРНК. В рамках настоящего изобретения можно использовать любую последовательность полиаденилирования, которая является функциональной в выбранной клетке-хозяине. Иллюстративные последовательности полиаденилирования для клеток-хозяев в виде нитчатых грибов включают, но не ограничиваются ими, последовательности полиаденилирования из генов амилазы TAKA Aspergillus oryzae, глюкоамилазы Aspergillus niger, антранилатсинтазы Aspergillus nidulans, трипсин-подобной протеазы Fusarium oxysporum и альфа-глюкозидазы Aspergillus niger. Пригодные последовательности полиаденилирования для дрожжевых клеток-хозяев также известны в данной области (см., например, Guo and Sherman, Mol. Cell. Bio., 15:5983-5990 [1995]).
[108] В некоторых вариантах осуществления последовательность контроля представляет собой кодирующую область сигнального пептида, которая кодирует аминокислотную последовательность, связанную с N-концом полипептида, и направляет кодируемый полипептид на секреторный путь клетки. 5'-конец кодирующей последовательности в последовательности нуклеиновой кислоты может по своей природе содержать область, кодирующую сигнальный пептид, естественным образом связанную в рамке считывания с сегментом кодирующей области, который кодирует секретируемый полипептид. Альтернативно 5'-конец кодирующей последовательности может содержать кодирующую область сигнального пептида, которая является чужеродной для кодирующей последовательности. Любая кодирующая область сигнального пептида, который направляет экспрессируемый полипептид на секреторный путь выбранной клетки-хозяина, является применимой для экспрессии сконструированных полипептидов РНК-полимеразы T7, описанных в настоящем описании. Эффективные кодирующие области сигнальных пептидов для клеток-хозяев в виде нитчатых дрожжей включают, но не ограничиваются ими, кодирующие области сигнальных пептидов, получаемые из генов амилазы TAKA Aspergillus oryzae, нейтральной амилазы Aspergillus niger, глюкоамилазы Aspergillus niger, аспарагиновой протеазы Rhizomucor miehei, целлюлазы Humicola insolens и липазы Humicola lanuginosa. Пригодные сигнальные пептиды для дрожжевых клеток-хозяев включают, но не ограничиваются ими, клетки-хозяева из генов альфа-фактора Saccharomyces cerevisiae и инвертазы Saccharomyces cerevisiae. Пригодные сигнальные пептиды для клеток-хозяев млекопитающих включают, но не ограничиваются ими, сигнальные пептиды из генов для иммуноглобулина-гамма (IgG).
[109] В некоторых вариантах осуществления последовательность контроля представляет собой область, кодирующую пропептид, которая кодирует аминокислотную последовательность, находящуюся на N-конце полипептида. Полученный пептид в некоторых случаях называют "проферментом", "прополипептидом" или "зимогеном". Прополипептид может конвертироваться в зрелый активный полипептид посредством каталитического или аутокаталитического отщепления пропептида от пропролипептида.
[110] В другом аспекте настоящее изобретение также относится к рекомбинантному экспрессирующему вектору, содержащему полинуклеотид, кодирующий сконструированный полипептид РНК-полимеразы T7, и одну или более областей, регулирующих экспрессию, таких как промотор и терминатор, ориджин репликации и т.д., в зависимости от типа хозяев, в которых его вводят. В некоторых вариантах осуществления различные последовательности нуклеиновых кислот и последовательности контроля, описанные выше, связаны вместе с получением рекомбинантного экспрессирующего вектора, который включает один или более удобных участков рестрикции для обеспечения инсерции или замещения последовательности нуклеиновой кислоты, кодирующей вариант полипептида РНК-полимеразы T7 в таких участках. Альтернативно последовательность(и) полинуклеотида по настоящему изобретению экспрессируют путем встраивания последовательности полинуклеотида или конструкции нуклеиновой кислоты, содержащей последовательность полинуклеотида в надлежащий вектор для экспрессии. При конструировании экспрессирующего вектора кодирующую последовательность размещают в векторе так, чтобы кодирующая последовательность была функционально связана с надлежащими последовательностями контроля для экспрессии.
[111] Рекомбинантный экспрессирующий вектор может представлять собой любой вектор (например, плазмиду или вирус), который удобно подвергать способам рекомбинантных ДНК и который может обеспечивать экспрессию варианта последовательности полинуклеотида РНК-полимеразы T7. Выбор вектора, как правило, зависит от совместимости вектора с клеткой-хозяином, в которую намереваются вводить вектор. Векторы могут представлять собой линейные или замкнутые кольцевые плазмиды.
[112] В некоторых вариантах осуществления экспрессирующий вектор представляет собой автономно реплицирующийся вектор (т.е. вектор, который существует в качестве внехромосомной структуры, репликация которой не зависит от хромосомной репликации, такой как плазмида, внехромосомный элемент, минихромосома или искусственная хромосома). Вектор может содержать любые средства для обеспечения саморепликации. В некоторых альтернативных вариантах осуществления вектор может представлять собой вектор, который при введении в клетку-хозяина встраивается в геном и реплицируется вместе с хромосомой(ами), в которую он встроился. Более того, можно использовать единый вектор или плазмиду или два или более векторов или плазмид, которые вместе содержат всю ДНК, подлежащую встраиванию в геном клетки-хозяина, или транспозон.
[113] В некоторых вариантах осуществления экспрессирующий вектор предпочтительно содержит один или более селективных маркеров, которые позволяют простую селекцию трансформированных клеток. "Селективный маркер" представляет собой ген, продукт которого обеспечивает резистентность к биоциду или вирусу, резистентность к тяжелым металлам, прототрофию ауксотрофам и т.п. Подходящие маркеры для дрожжевых клеток включают, но не ограничиваются ими, ADE2, HIS3, LEU2, LYS2, MET3, TRP1 и URA3. Селективные маркеры для применения в клетках-хозяевах в виде нитчатых грибов включают, но не ограничиваются ими, amdS (ацетамидаза), argB (орнитинкарбамоилтрансферазы), bar (фосфинотрицинацетилтрансфераза), hph (гигромицинфосфотрансфераза), niaD (нитратредуктаза), pyrG (оротидин-5'-фосфатдекарбоксилаза), sC (сульфатаденилтрансфераза) и trpC (антранилатсинтаза), а также их эквиваленты. В другом аспекте настоящее изобретение относится клетке-хозяину, содержащей полинуклеотид, кодирующий по меньшей мере один сконструированный полипептид РНК-полимеразы T7, описанный в настоящей заявке, причем полинуклеотид функционально связан с одной или более последовательностями контроля для экспрессии сконструированного фермента(ов) РНК-полимеразы T7 в клетке-хозяине. Клетки-хозяева для экспрессии полипептидов, кодируемых экспрессирующими векторами по настоящему изобретению, хорошо известны в данной области и включают, но не ограничиваются ими, клетки грибов, такие как дрожжевые клетки (например, Saccharomyces cerevisiae и Pichia pastoris [например, номер доступа ATCC №201178]); клетки насекомых (например, клетки Drosophila S2 и Spodoptera Sf9), клетки растений, клетки животных (например, CHO, COS и BHK) и клетки человека (например, клеточные линии HEK293T, фибробластов человека, THP-1, Jurkat и меланомы Bowes).
[114] Таким образом, в другом аспекте настоящее изобретение относится к способам получения сконструированных полипептидов РНК-полимеразы T7, где способы включают культивирование клетки-хозяина, способной экспрессировать полинуклеотид, кодирующий сконструированный полипептид РНК-полимеразы T7, в условиях, пригодных для экспрессии полипептида. В некоторых вариантах осуществления способы дополнительно включают стадии выделения и/или очистки полипептидов РНК-полимеразы T7, как описано в настоящем описании.
[115] Надлежащие культуральные среды и условия выращивания описанных выше клеток-хозяев хорошо известны в данной области. Полинуклеотиды для экспрессии полипептидов РНК-полимеразы T7 можно вводить в клетки различными способами, известными в данной области. Способы включают, среди прочих, электропорацию, бомбардировку биолистическими частицами, опосредуемую липосомами трансфекцию, трансфекцию с хлоридом кальция и слияние протопластов.
[116] Сконструированную РНК-полимеразу T7 со свойствами, описанными в настоящим описании, можно получать, подвергая полинуклеотид, кодирующий встречающийся в природе или сконструированный полипептид РНК-полимеразы T7, способам мутагенеза и/или направленной эволюции, известным в данной области, и как описано в настоящем описании. Иллюстративным способом направленной эволюции является мутагенез и/или шаффлинг ДНК (см., например, Stemmer, Proc. Natl. Acad. Sci. USA 91:10747-10751 [1994]; WO 95/22625; WO 97/0078; WO 97/35966; WO 98/27230; WO 00/42651; WO 01/75767 и патент США 6537746). Другие способы направленной эволюции, которые можно использовать, включают, среди прочих, способ ступенчатой достройки (StEP), рекомбинацию in vitro (см., например, Zhao et al., Nat. Biotechnol., 16:258-261 [1998]), мутагенную ПЦР (см., например, Caldwell et al., PCR Methods Appl., 3:S136-S140 [1994]) и кассетный мутагенез (см., например, Black et al., Proc. Natl. Acad. Sci. USA 93:3525-3529 [1996]).
[117] Например, способы мутагенеза и направленной эволюции можно без труда применять в отношении полинуклеотидов для получения вариантов библиотек, которые можно экспрессировать, подвергать скринингу и оценивать. Способы мутагенеза и направленной эволюции хорошо известны в данной области (см., например, патенты США №5605793, 5811238, 5830721, 5834252, 5837458, 5928905, 6096548, 6117679, 6132970, 6165793, 6180406, 6251674, 6277638, 6287861, 6287862, 6291242, 6297053, 6303344, 6309883, 6319713, 6319714, 6323030, 6326204, 6335160, 6335198, 6344356, 6352859, 6355484, 6358740, 6358742, 6365377, 6365408, 6368861, 6372497, 6376246, 6379964, 6387702, 6391552, 6391640, 6395547, 6406855, 6406910, 6413745, 6413774, 6420175, 6423542, 6426224, 6436675, 6444468, 6455253, 6479652, 6482647, 6489146, 6506602, 6506603, 6519065, 6521453, 6528311, 6537746, 6573098, 6576467, 6579678, 6586182, 6602986, 6613514, 6653072, 6716631, 6777218, 6917882, 6946296, 6961664, 6995017, 7024312, 7058515, 7105297, 7148054, 7288375, 7421347, 7430477, 7534564, 7620500, 7620502, 7629170, 7702464, 7747391, 7747393, 7751986, 7776598, 7783428, 7795030, 7853410, 7868138, 7873477, 7873499, 7904249, 7957912, 8014961, 8029988, 8058001, 8076138, 8018150, 8170806, 8377681, 8383346, 8457903, 8504498, 8589085, 8762066, 8849575, 8876066, 8768871, 9593326, 9665694 и все родственные аналоги, происходящие из США и не США; Ling et al., Anal. Biochem., 254(2):157-78 [1997]; Dale et al., Meth. Mol. Biol., 57:369-74 [1996]; Smith, Ann. Rev. Genet., 19:423-462 [1985]; Botstein et al., Science, 229:1193-1201 [1985]; Carter, Biochem. J., 237:1-7 [1986]; Kramer et al., Cell, 38:879-887 [1984]; Wells et al., Gene, 34:315-323 [1985]; Minshull et al., Curr. Op. Chem. Biol., 3:284-290 [1999]; Christians et al., Nat. Biotechnol., 17:259-264 [1999]; Crameri et al., Nature, 391:288-291 [1998]; Crameri, et al., Nat. Biotechnol., 15:436-438 [1997]; Zhang et al., Proc. Nat. Acad. Sci. U.S.A., 94:4504-4509 [1997]; Crameri et al., Nat. Biotechnol., 14:315-319 [1996]; Stemmer, Nature, 370:389-391 [1994]; Stemmer, Proc. Nat. Acad. Sci. USA, 91:10747-10751 [1994]; публикации патентных заявок США №2008/0220990, US 2009/0312196, US 2014/0005057, US 2014/0214391, US 2014/0221216; US 2015/0050658, US 2015/0133307, US 2015/0134315 и все родственные аналоги, происходящие из США и не США; WO 95/22625, WO 97/0078, WO 97/35966, WO 98/27230, WO 00/42651, WO 01/75767 и WO 2009/152336; все из которых включены в настоящее описание в качестве ссылок).
[118] В некоторых вариантах осуществления варианты ферментов, полученные после мутагенной обработки, подвергают скринингу, подвергая варианты ферментов воздействию определенной температуры (или других условиях анализа) и измеряя величину ферментативной активности после термической обработки или других условий анализа. Затем ДНК, содержащую полинуклеотид, кодирующий полипептид РНК-полимеразы T7, выделяют из клетки-хозяина, секвенируют для идентификации изменений нуклеотидной последовательности (при наличии) и используют для экспрессии фермента в отличающейся или той же клетке-хозяине. Определение ферментативной активности в экспрессирующих библиотеках можно проводить с использованием любого подходящего способа, известного в данной области (например, стандартные способы биохимии, таки как анализ с использованием ВЭЖХ).
[119] Для сконструированных полипептидов известной последовательности полинуклеотиды, кодирующие фермент, можно получать стандартными твердофазными способами в соответствии с известными способами синтеза. В некоторых вариантах осуществления фрагменты длиной вплоть до приблизительно 100 оснований можно синтезировать по отдельности, а затем соединять (например, способами ферментативного или химического лигирования или опосредуемыми полимеразами способами) с получением любой желаемой непрерывной последовательности. Например, полинуклеотиды и олигонуклеотиды, описанные в настоящем описании, можно получать посредством химического синтеза с использованием классического способа с фосфорамидитом (cм., например, Beaucage et al., Tetra. Lett., 22:1859-69 [1981]; и Matthes et al., EMBO J., 3:801-05 [1984]), поскольку он обычно применяется на практике в автоматизированных способах синтеза. В соответствии со способом с фосфорамидитом олигонуклеотиды синтезируют (например, в автоматизированном устройстве для синтеза ДНК), очищают, подвергают отжигу, лигируют и клонируют в подходящие векторы.
[120] Таким образом, в некоторых вариантах осуществления способ получения сконструированного полипептида РНК-полимеразы T7 может включать: (a) синтез полинуклеотида, кодирующего полипептид, содержащий аминокислотную последовательность, выбранную из аминокислотной последовательности любого варианта, приведенного в таблице 5.3, 5.4, 5.5 и/или 5.6, а также SEQ ID NO: 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37 и 39, и (b) экспрессию полипептида РНК-полимеразы T7, кодируемого полинуклеотидом. В некоторых вариантах осуществления способа аминокислотная последовательность, кодируемая полинуклеотидом, необязательно может иметь одну или более (например, вплоть до 3, 4, 5 или вплоть 10) делеций, инсерций и/или замен аминокислотных остатков. В некоторых вариантах осуществления аминокислотная последовательность необязательно имеет 1-2, 1-3, 1-4, 1-5, 1-6, 1-7, 1-8, 1-9, 1-10, 1-15, 1-20, 1-21, 1-22, 1-23, 1-24, 1-25, 1-30, 1-35, 1-40, 1-45 или 1-50 делеций, инсерций и/или замен аминокислотных остатков. В некоторых вариантах осуществления аминокислотная последовательность имеет необязательно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 30, 30, 35, 40, 45 или 50 делеций, инсерций и/или замен аминокислотных остатков. В некоторых вариантах осуществления аминокислотная последовательность имеет необязательно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 18, 20, 21, 22, 23, 24 или 25 делеций, инсерций и/или замен аминокислотных остатков. В некоторых вариантах осуществления замены могут представлять собой консервативные или неконсервативные замены.
[121] Экспрессируемый сконструированный полипептид РНК-полимеразы T7 можно оценивать в отношении любого желаемого усовершенствованного свойства (например, активность, селективность, стабильность, устойчивость к действию кислот, чувствительность к протеазам и т.д.), с использованием любого подходящего способа анализа, известного в данной области, включая, но не ограничиваясь ими способы анализа и условия, описанные в настоящем описании.
[122] В некоторых вариантах осуществления любой из сконструированных полипептидов РНК-полимеразы T7, экспрессируемых в клетке-хозяине, выделяют из клеток и/или культуральной среды с использованием любого одного или более из хорошо известных способов очистки белков, включая, среди прочих, обработку лизоцимом, обработку ультразвуком, фильтрацию, высаливание, ультрацентрифугирование и хроматографию.
[123] Хроматографические способы выделения полипептидов T7 РНК-полимеразы включают, среди прочих, обращено-фазовую хроматографию, высокоэффективную жидкостную хроматографию, ионообменную хроматографию, хроматографию гидрофобного взаимодействия, гель-электрофорез и аффинную хроматографию. Условия для очистки конкретных ферментов зависят, частично, от таких факторов, как суммарный заряд, гидрофобность, гидрофильность, молекулярная масса, молекулярная форма и т.д., и станут понятными специалистам в данной области. В некоторых вариантах осуществления аффинные способы можно использовать для выделения усовершенствованных вариантов ферментов РНК-полимеразы. В некоторых вариантах осуществления, в которых используется очистка посредством аффинной хроматографии, может использоваться любое антитело, которое специфически связывает вариант полипептида РНК-полимеразы T7. В некоторых вариантах осуществления, в которых используется очистка посредством аффинной хроматографии, могут использоваться белки, которые связываются с гликанами, ковалентно связанными с РНК-полимеразой T7. В других вариантах осуществления, в которых используется очистка посредством аффинной хроматографии, может использоваться любое низкомолекулярное соединение, которое связывается с активным центром РНК-полимеразы T7. Для продуцирования антител различных животных-хозяев, включая, но не ограничиваясь ими, кроликов, мышей, крыс и т.д., иммунизируют посредством инъекции полипептида РНК-полимеразы T7 (например, варианта РНК-полимеразы T7) или его фрагмента. В некоторых вариантах осуществления полипептид РНК-полимераза T7 или его фрагмент связан с подходящим носителем, таким как BSA, через функциональную группу боковой цепи или линкеры, связанные с функциональной группой боковой цепи.
[124] В некоторых вариантах осуществления сконструированный полипептид РНК-полимеразы T7 продуцируют в клетке-хозяине способом, включающим культивирование клетки-хозяина (например, S. cerevisiae, Daucus carota, Nicotiana tabacum, H. sapiens (например, HEK293T) или Cricetulus griseus (например, CHO)), содержащей последовательность полинуклеотида, кодирующую сконструированный полипептид РНК-полимеразы T7, как описано в настоящем описании, в условиях, способствующих продуцированию сконструированного полипептида РНК-полимеразы T7, и выделение сконструированного полипептида РНК-полимеразы T7 из клеток и/или культуральной среды.
[125] В некоторых вариантах осуществления изобретение охватывает способ продуцирования сконструированного полипептида РНК-полимеразы T7, включающий культивирование рекомбинантной эукариотической клетки, содержащей последовательность полинуклеотида, кодирующего сконструированный полипептид РНК-полимеразы T7, обладающую по меньшей мере 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 95%, 96%, 97%, 98%, 99% или 100% идентичностью последовательности с эталонными последовательностями SEQ ID NO: 4 и/или 15, и одно или более отличий аминокислотных остатков по сравнению с SEQ ID NO: 4 и/или 15, выбранных из различий, приведенных в таблицах 5.3, 5.4, 5.5 и/или 5.6, и/или их комбинаций при оптимальном выравнивании с аминокислотной последовательностью SEQ ID NO: 4 и/или 15 в подходящих условиях культивирования для обеспечения продуцирования сконструированного полипептида РНК-полимеразы T7, и необязательно выделение сконструированного полипептида РНК-полимеразы T7 из культуры и/или культивируемых бактериальных клеток.
[126] В некоторых вариантах осуществления после выделения сконструированных полипептидов РНК-полимеразы T7 из рекомбинантных клеток-хозяев или среды для культивирования клеток, их далее очищают любым подходящим способом(ами), известным в данной области. В некоторых дополнительных вариантах осуществления очищенные полипептиды РНК-полимеразы T7 комбинируют с другими ингредиентами и соединениями для обеспечения композиций и составов, содержащих сконструированный полипептид РНК-полимеразы T7, надлежащим образом для различных способов использования и применений (например, фармацевтические композиции). В некоторых дополнительных вариантах осуществления очищенные полипептиды РНК-полимеразы T7 или составленные полипептиды РНК-полимеразы T7 являются лиофилизированными.
Композиции:
[127] Настоящее изобретение относится к различным композициям и форматам, включая, но не ограничиваясь ими, композиции и форматы, описанные ниже. В некоторых вариантах осуществления настоящее изобретение относится сконструированным полипептидам РНК-полимеразы T7, пригодным для применения в композициях для диагностических целей.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
[128] Следующие примеры, включая эксперименты и достигнутые результаты, предоставлены только для иллюстративных целей, и их не следует истолковывать как ограничивающие настоящее изобретение.
[129] В описании экспериментальной части ниже используются следующие сокращенные обозначения: м.д. (части на миллион); M (молярный); мМ (миллимолярный), мкМ (микромолярный); нМ (наномолярный); моль (количество моль); г (граммы); мг (миллиграммы); мкг (микрограммы); л (литры); мл (миллилитры); см (сантиметры); мм (миллиметры); мкм (микрометры); с (секунды); мин (минута(ы)); ч (час(ы)); Е (единицы); ММ (молекулярная масса); об/мин (обороты в минуту); rcf (относительная центробежная сила); °C (градусы Цельсия); CDS (кодирующая последовательность); ДНК (дезоксирибонуклеиновая кислота); РНК (рибонуклеиновая кислота); E. coli W3110 (часто используемый лабораторный штамм E. coli, доступный от Coli Genetic Stock Center [CGSC], New Haven, CT); ВЭЖХ (жидкостная хроматография высокого давления); MWCO (предел молекулярной массы); SDS-PAGE (полиакриламидный гель-электрофорез с додецилсульфатом натрия); T7RNAP (РНК-полимераза T7); PES (полиэфирсульфон); CFSE (сукцинимидиловый эфир карбоксифлуоресцеина); IPTG (изопропил β-D-1-тиогалактопиранозид); PMBS (сульфат полимиксина B); NADPH (никотинамидадениндинуклеотидфосфат); GIDH (глутаматдегидрогеназа); FIOPC (кратность улучшения относительно положительного контроля); LB (бульон Луриа); MeOH (метанол); Athens Research (Athens Research Technology, Athens, GA); NEB (New England Biolabs, Ipswich, MA); Ion Torrent (Ion Torrent, Gilford, NH); ProSpec (ProSpec Tany Technogene, East Brunswick, NJ); Sigma-Aldrich (Sigma-Aldrich, St. Louis, MO); Ram Scientific (Ram Scientific, Inc., Yonkers, NY); Pall Corp. (Pall, Corp., Pt. Washington, NY); Millipore (Millipore, Corp., Billerica MA); Difco (Difco Laboratories, BD Diagnostic Systems, Detroit, MI); Molecular Devices (Molecular Devices, LLC, Sunnyvale, CA); Kuhner (Adolf Kuhner, AG, Basel, Switzerland); Axygen (Axygen, Inc., Union City, CA); Toronto Research Chemicals (Toronto Research Chemicals Inc., Toronto, Ontario, Canada); Cambridge Isotope Laboratories, (Cambridge Isotope Laboratories, Inc., Tewksbury, MA); Applied Biosystems (Applied Biosystems, part of Life Technologies, Corp., Grand Island, NY), Agilent (Agilent Technologies, Inc., Santa Clara, CA); Thermo Scientific (part of Thermo Fisher Scientific, Waltham, MA); Ion Torrent NGS (Ion Torrent Next Generation Sequencing); Li-COR (Li-COR, Lincoln, NE); UVP (UVP, Upland, CA); Biotium (Biotium, Inc., Fremont, CA); Corning (Corning, Inc., Palo Alto, CA); Megazyme (Megazyme International, Wicklow, Ireland); Enzo (Enzo Life Sciences, Inc., Farmingdale, NY); GE Healthcare (GE Healthcare Bio-Sciences, Piscataway, NJ); Pierce (Pierce Biotechnology (now part of Thermo Fisher Scientific), Rockford, IL); LI-COR (LI-COR Biotechnology, Lincoln, NE); Amicus (Amicus Therapeutics, Cranbury, NJ); Phenomenex (Phenomenex, Inc., Torrance, CA); Optimal (Optimal Biotech Group, Belmont, CA); и Bio-Rad (Bio-Rad Laboratories, Hercules, CA).
[130] Следующие полинуклеотидные или полипептидные последовательности применимы в рамках настоящего изобретения. В некоторых случаях (как показано ниже) после полинуклеотидной последовательности следует кодируемый полипептид.
ПРИМЕР 1
Получение гена РНК-полимеразы T7 и конструирование экспрессирующих векторов
[131] Фермент РНК-полимераза T7 дикого типа (WT) (SEQ ID NO: 2) кодируется геномом бактериофага T7 (SEQ ID NO: 1). Синтетический ген (SEQ ID NO: 3), кодирующий меченную 6-гистидином версию РНК-полимеразы T7 (SEQ ID NO: 4), конструировали и субклонировали в экспрессирующий вектор Escherichia coli pCK100900i (см., например, патент США №7629157 и публикацию патентной заявки США №2016/0244787, обе из которых включены в настоящее описание в качестве ссылок). Эти плазмидные конструкции трансформировали в штамм E. Coli, происходящий из W3110. Способы направленной эволюции, общеизвестные специалистам в данной области, использовали для получения библиотек вариантов генов из этих плазмид (см., например, патент США №8383346 и WO 2010/144103, оба из которых включены в настоящее описание в качестве ссылок). Замены в вариантах ферментов, описанных в настоящем описании, указаны относительно фермента 6His-меченной РНК-полимеразы T7 WT (т.е. SEQ ID NO: 4) или их вариантов, как указано.
ПРИМЕР 2
Высокопроизводительная (HTP) экспрессия и очистка РНК-полимеразы T7
[132] В этом примере описаны эксперименты, проведенные в отношении экспрессии и очистки РНК-полимеразы T7 и ее вариантов.
Высокопроизводительное (HTP) выращивание ДНК-полимеразы T7 и вариантов
[133] Трансформированные клетки E. coli подвергали селекции путем посева на чашки с агаром LB, содержавшие 1% глюкозу и 30 мкг/мл хлорамфеникола. После инкубации в течение ночи при 37°C, колонии помещали в лунки 96-луночных неглубоких планшетов с плоским дном NUNC™ (Thermo-Scientific), заполненных 180 мкл/лунка средой LB, дополненной 1% глюкозой и 30 мкг/мл хлорамфеникола. Культурам позволяли расти ночью в течение 18-20 часов в устройстве для встряхивания (200 об/мин, 30°C и относительная влажность 85%; Kuhner). Образцы, подвергнутые выращиванию в течение ночи (20 мкл), переносили в 96-луночные глубоки планшеты Costar, заполненные 380 мкл среды Terrific Broth, дополненной 30 мкг/мл хлорамфеникола. Планшеты инкубировали в течение 120 минут в устройстве для встряхивания (250 об/мин, 30°C и относительная влажность 85%; Kuhner) до достижения OD600 0,4-0,8. Затем клетки индуцировали посредством 40 мкл 10 мМ IPTG в стерильной воде и инкубировали в течение ночи в течение 18-20 часов в устройстве для встряхивания (250 об/мин, 30°C и относительная влажность 85%; Kuhner). Клетки осаждали (4000 об/мин×20 мин), супернатанты удаляли и клетки замораживали при -80°C перед анализом.
Лизис осадков HTP
[134] Клеточные осадки ресуспендировали в 400 мкл лизирующего буфера (20 мМ Tris, pH 7,5, 1 MgSO4, 1 мг/мл лизоцима и 0,5 мг/мл сульфата полимиксина B) и встряхивали в течение 1,5 ч при комнатной температуре. Затем лизаты осаждали (4000 об/мин×5 мин) и отделенные от примесей супернатанты сохраняли для очистки.
Очистка HTP T7RNAP из неочищенных лизатов
[135] T7RNAP очищали из отделенных от примесей лизатов E. coli посредством металл-аффинной хроматографии с использованием покрытых никелем планшетов HIS-Select® большого объема (HC) (Sigma) в соответствии с инструкциями изготовителя. Планшеты HIS-Select® уравновешивали всего 800 мкл промывочного буфера (50 мМ фосфат натрия, pH 7,5, 300 мМ NaCl, 25 мМ имидазол, 0,1% об./об. реагента TWEEN-20® [Sigma]) на лунку. Затем 200 мкл лизата HTP, содержавшего T7RNAP, и 200 мкл промывочного буфера перемешивали, помещали в планшет и центрифугировали в течение 1 мин при относительной центробежной силе 2000 (rcf) и 4°C. Планшет промывали два раза 600 мкл промывочного буфера на лунку с центрифугированием в течение 3 мин при 3000 rcf и 4°C для каждого промывания. Образцы ферментов элюировали добавлением 200 мкл элюирующего буфера (50 мМ фосфат натрия, pH 7,5, 300 мМ NaCl, 250 мМ имидазола, 0,1% об./об. реагента TWEEN®-20) посредством центрифугирования в течение 1 мин при 3000 rcf при 4°C.
[136] Элюаты подвергали замене буфера с использованием обессоливающих планшетов Zeba™ Spin (Thermo Fisher). В кратком изложении, планшеты уравновешивали два раза посредством 375 мкл 2× буфера для хранения T7RNAP (100 мМ Tris.HCl, pH 8,0, 200 мМ NaCl, 2 мМ DTT, 2 мМ EDTA, 0,2% масс./об. Triton X-100) на лукну и центрифугировали в течение 2 мин при 1100 xg при 4°C. В обессоливающие планшеты помещали 100 мкл элюата с планшета для образца HIS-Select® и центрифугировали в течение 2 мин при 1100 rcf при 4°C. Элюат с обессоливающего планшета сохраняли и смешивали с равным объемом глицерина для конечной концентрации буфера для хранения, содержавшего 50 мМ Tris HCl pH 8,0, 100 мМ NaCl, 1 мМ DTT, 1 мМ EDTA, 0,1% об./об. Triton X-100 и 50% глицерин (об./об.).
[137] Отсутствие контаминации РНК-азой в очищенных образцах подтверждали с использованием анализа RNase Alert (IDT, Life Technologies). Анализ SDS-PAGE для образцов T7RNAP продемонстрировал отсутствие поддающихся обнаружению соответствующих контаминации полос для большинства образцов.
[138] Для реакций транскрипции in vitro с использованием HTP-очищенной T7RNAP, очищенную полимеразу добавляли до конечной концентрации 10% общего объема реакции (5% для фермента из культур во вращающихся флаконах).
ПРИМЕР 3
Экспрессия во вращающихся флаконах и очистка РНК-полимеразы T7
[139] В этом примере описаны эксперименты, вовлекающие экспрессию во вращающихся флаконах и очистку T7RNAP.
Экспрессия во вращающихся флаконах
[140] Отобранные культуры HTP, выращенные как описано в примере 2, высевали на чашки с агаром LB с 1% глюкозой и 30 мкг/мл хлорамфеникола и выращивали в течение ночи при 37°C. Единичную колонию из каждой культуры переносили в 6 мл бульона LB с 1% глюкозой и 30 мкг/мл хлорамфеникола. Культуры выращивали в течение 18 ч при 30°C, 250 об/мин и субкультивировали при разведении приблизительно 1:10 в 250 мл среды Terrific Broth с 30 мкг/мл хлорамфеникола до конечной OD600 0,2. Культуры инкубировали в течение приблизительно 3 часов при 30°C, 250 об/мин, до OD600 0,6-0,8, а затем индуцировали добавлением IPTG в конечной концентрации 1 мМ. Индуцированные культуры инкубировали в течение 20 ч при 30°C, 250 об/мин. После этого периода инкубации культуры центрифугировали при 4000 об/мин×10 мин. Культуральный супернатант удаляли и осадки ресуспендировали в 35 мл лизирующего буфера (50 мМ NaH2PO4, pH 7,5, 500 мМ NaCl, 0,1% Tween-20, 10 мМ имидазол). Эту клеточную суспензию охлаждали на ледяной бане и лизировали с использованием устройства для разрушения клеток Microfluidizer (Microfluidics M‐110L). Неочищенный лизат осаждали центрифугированием (16000 об/мин в течение 60 мин при 4°C), а затем супернатант фильтровали через 0,2-мкм мембрану PES для дальнейшей очистки лизата.
Очистка РНК-полимеразы T7 из лизатов из вращающихся флаконов
[141] Лизаты T7RNAP очищали с использованием системы для очистки AKTA Start и 5-мл колонки HisTrap FF (GE Healthcare) с использованием условий AC Step HiF (рабочие параметры приведены ниже). Промывочный буфер SF содержал 50 мМ фосфат натрия, pH 7,5, 500 мМ NaCl, 0,1% об./об. реагента TWEEN-20® (Sigma) и 25 мМ имидазол. Элюирующий буфер SF содержал 50 мМ фосфат натрия, pH 7,5, 500 мМ NaCl, 0,1% об./об. реагента TWEEN-20® (Sigma) и 300 мМ имидазол.
[142] Пять единичных наиболее концентрированных 3-мл фракций идентифицировали посредством УФ-поглощения (A280) и подвергали диализу в течение ночи в 2× буфере для хранения T7RNAP (100 мМ Tris HCl pH 8,0, 200 мМ NaCl, 2 мМ DTT, 2 мМ EDTA, 0,2% об./об. Triton X-100) в течение ночи в кассете для диализа 10K Slide-A-Lyzer™ (Thermo Fisher) для замены буфера в течение 16 часов, за которой следовала вторая замена буфера на 24 часа. К подвергнутому диализу материалу добавляли равный объем глицерина. Концентрации ферментов в препаратах количественно определяли посредством денситометрии на геле и по поглощению при 280 нм.
ПРИМЕР 4
Реакции транскрипции
[143] Реакции транскрипции проводили с аналогом кэпа альфа, гамма-бис(N7-метилгуанозин)трифосфатом (также обозначаемым в настоящем описании как "m7G(5')ppp(5')m7G," "кэпированный GTP" или "Cap") (см., Grudzien et. al., RNA, 10:1479-87 [2004]). Сконструированная ДНК-матрица для транскрипции GlmS-16A (SEQ ID NO: 5) включает промоторную последовательность T7RNAP, связанную с кодирующей последовательностью для рибопереключателя GlmS Bacillus anthracis (SEQ ID NO: 6). При индукции посредством его лиганда глюкозамин-6-фосфата рибопереключатель GlmS подвергается саморасщеплению и высвобождает 16-мерный РНК-нуклеотид (SEQ ID NO: 7) с 5'-конца транскрипта, который включает структуру кэпа или некэпированный 5'-фосфат. Этот небольшой 16-мерный продукт расщепления РНК-олигонуклеотида подвергали ионизации и анализу посредством LC-MS, который использовали, чтобы отличить кэпированные и некэпированные типы.
[144] Реакции (30 мкл) проводили с конечными концентрациями 50 мМ Tris HCl pH 7,9, 30 мМ MgCl2, 10 мМ DTT, 6 мМ ATP, 6 мМ CTP, 6 мМ UTP, 4,8 мМ GTP, 1,2 мМ m7G(5')ppp(5')m7G, 50 нг/мкл матрицы транкрипции GlmS-16A, 1 Е/мкл ингибитора RNasin, и 6,4 мМ глюкозамин-6-фосфат. Глюкозамин-6-фосфат включали в начале реакции и расщеплению рибопереключателя позволяли протекать в ходе транскрипции in vitro в течение 4 часов. Для HTP-скрининга реакционные смеси гасили равным (30 мкл) объемом 40 мМ EDTA. Общее количество m7G(5')ppp(5')m7G и GTP поддерживали на уровне 6 мМ.
ПРИМЕР 5
Анализ активности LC-MS
[145] В этом примере описан способ LC-MS, разработанный для разделения фрагментов РНК меньшего размера с использованием системы Thermo LTQ MS для анализа более коротких кэпированных 5'- и некэпированных 5'-трифосфатных продуктов расщепления.
[146] Кэпированные 7meG и 5'-трифосфатные некэпированные 16-мерные продукты расщепления (SEQ ID NO: 7) подвергали хроматографическому разделению (cм. фиг.1) от реагентов и более высокомолекулярных РНК с использованием способа обращено-фазовой ВЭЖХ с образованием ионных пар с использованием колонки C18 и подвижной фазы HFIP, обычно используемой для разделения олигонуклеотидов посредством масс-спектрометрии (см. таблицу 5.1).
Таблица 5.1 Способ LC-MS, использованный для детекции кэпированных и некэпированных 16-мерных продуктов расщепления
(взвесить HFIP, медленно растворить добавляемую TEA)
B: 200 мМ HFIP, ~8,15 мМ TEA в смеси вода/метанол 50/50 (использовать A для получения B)
Экстрагированные ионы BP для:
- Некэпированного 16-мерного продукта=903,8, 1084,6, 1355,9, 1361,4, 1808,1, 1815,6, 1820,8
- Кэпированного 16-мерного продукта=952,7, 1143,4, 1429,2, 1438,7, 1906,1, 1918,6, 1931,3
[147] Определение основных пиков для кэпированных и некэпированных 16-мерных продуктов расщепления проводили с использованием семи ионов для каждого типа, включая множество отрицательных заряженных ионов и аддуктов металлов. Интенсивность пика для этих семи ионов использовали для получения сигнала для каждого из кэпированных и некэпированных 16-мерных продуктов расщепления. Эти оны включают множество заряженных состояний, а также аддукты натрия и калия (см. таблицу 5.2). Все ионы наблюдали в пределах 1 единицы массы от ожидаемых величин.
Таблица 5.2 Ионы, подвергнутые определению для масс-спектрометрической интеграции
[148] Эффективность кэпирования в отношении WT T7RNAP (т.е "кратность улучшения относительно исходной молекулы" или "FIOP") вычисляли путем деления отношений интенсивности пиков кэпированных/некэпированных молекул для каждого образца на отношения, вычисленные для исходных контролей WT, присутствующих в каждом планшете (n=6-10). Величину FIOP использовали для ранжирования эффективности вариантов относительно WT T7RNAP или исходной молекулы из библиотеки, которые были включены в каждый планшет в качестве контролей. С использованием этого способа относительного количественного определения было возможным нивелирование различий в ионизации, которые присущи кэпированным и некэпированным молекулам, а также вариаций сигнала, обеспечиваемых партией подвижной фазы HFIP и концентрацией EDTA.
Реакции транскрипции in vitro проводили, как описано в примере 4, с временем реакции и концентрациями m7G(5')ppp(5')m7G, как указано в таблицах, которые приведены ниже. В таблице 5.3, 5.4 и 5.5 представлено среднее увеличение активности для 3-6 реплик. В таблице 5.6 представлено среднее улучшение активности для вариантов, где была доступна информация для реплик.
Таблица 5.3 Увеличение активности вариантов T7RNAP
Таблица 5.4 Увеличение активности варианта T7RNAP
[149] Отмечалось, что вариант 85 имел не поддающиеся обнаружению уровни некэпированной мРНК в анализе LC-MS.
Таблица 5.5 Увеличение активности варианта T7RNAP
Таблица 5.6 Увеличение активности варианта T7RNAP
SEQ ID NO: 4
+++ = ≥15
++ = 7-14,9
+ = 2-6,9
ПРИМЕР 6
Определение выхода РНК
[150] Реакции транскрипции in vitro проводили, как описано в примере 4, но с использованием матрицы люциферазы (SEQ ID NO: 10). Выход мРНК из реакций транскрипции in vitro определяли в соответствии с протоколом изготовителя набора Quant-iT РНК Assay kit (широкий диапазон, Q-33140, Thermo Fisher). Содержание мРНК вычисляли с использованием стандартной кривой, построенной для стандартов мРНК, предоставленных с набором для анализа.
++ = > выход РНК 1,8 мг/мл,
ПРИМЕР 7
Определение точности транскрипции варианта T7RNAP
[151] Точность полимеразы определяли на основе прямого секвенирования большого количества клонов ОТ-ПЦР, полученных из мРНК, транскрибированной с варианта полимеразы. Реакции транскрипции in vitro проводили, как описано в примере 4, с использованием 0,5 мМ m7G(5')ppp(5')m7G, 5,5 мМ GTP и без использования глюкозамин-6-фосфата. Включение m7G(5')ppp(5')m7G позволяло количественное определение вклада этого аналога кэпа (при его наличии) в уровень погрешности для WT и вариантов полимераз в связанных со способом условиях. ДНК-матрица люциферазы размером 1,7 т.п.н. (SEQ ID NO: 8) служила в качестве ДНК-матрицы для транскрипции с использованием RNAP T7 дикого типа и вариантов RNAP T7 для получения полноразмерных мРНК-транскриптов. РНК выделяли с использованием набора Zymo RNA Clean and concentrator-25 (Zymo Research) и остаточную ДНК удаляли из образцов РНК посредством двух последовательных обработок набором DNA-free DNAase I (Ambion/ Thermo Fisher). Образцы подвергали обратной транскрипции и использованием обратной транскриптазы Accuprime (Agilent) с использованием олиго-(dT)25 праймера (SEQ ID NO: 40), гибридизующегося с поли(A)-хвостовой частью на матрице люциферазы. Затем реакционную смесь RT подвергали амплификации с использованием высокоточной ДНК-полимеразы PHUSION® с использованием буфера HF (New England Biolabs) посредством ПЦР с получением ампликона размером 1675 п.н. с использованием специфических для гена праймеров (SEQ ID NO: 12, SEQ ID NO: 13), гибридизующихся с кодирующей последовательностью люциферазы. Амплифицированные фрагменты расщепляли посредством BglI (New England Biolabs), лигировали в клонирующий вектор и трансформировали с получением единичных клонов в E. coli.
[152] Индивидуальные клоны отбирали и секвенировали с использованием стратегии мультиплексного баркодирования на платформе Ion Torrent PGM (Thermo Fisher). Баркодированные считанные последовательности подвергали обратной свертке, а затем последовательности индивидуальных клонов вновь собирали против предполагаемой последовательности матрицы для области размером 1632 п.н. между SEQ ID NO: 12 и SEQ ID NO: 13. Мутации, включая небольшие инсерции, делеции (т.е. инсерции-делеции) и однонуклеотидные полиморфизмы, регистрировали. Большинство мутаций представляли собой замены, хотя также наблюдались инсерции и делеции. Общее количество мутаций на пару оснований отсеквенированных происходящих из мРНК клонов вычисляли, и оно представлено в таблицах 7.1 и 7.2. Ожидаемые общие частоты мутаций на пару оснований, исходя из литературных данных, представляют собой сумму ошибок вследствие T7RNAP (1×10-4) (Huang et al., Biochem., 39:11571-11580 [2000]), обратной транскриптазы Accuscript (6×10-5) (Agilent; См., product literature) и ДНК-полимеразы Phusion (1,2×10-5) (20 циклов; см. литературу о продукте NEB) или в общем 1,72×10-4. Большинство вариантов полимераз продемонстрировали показатели ошибок, близкие к описанной в литературе величине для T7RNAP, даже в присутствии 0,5 мМ m7G(5')ppp(5')m7G.
[153] Односторонний (правосторонний) биноминальный критерий использовали для вычисления случайности выборки для наблюдаемого количества ошибок (или более), учитывая количество отсеквенированных оснований, если фактический уровень ошибки в эксперименте равен наблюдаемой общей частоте ошибок для T7RNAP-WT в отдельных экспериментах, представленных в таблицах 7.1 и 7.2. Точность данного варианта считалась неотличимой от WT T7RNAP в этом анализе для величин p более 0,05. В таблице 7.1 "+" составляет менее 1,7*10-4, и "-" превышает 1,7*10-4 в столбце результата "наблюдаемая частота ошибок", в то время как "+" представляет собой p > 0,05 и "-" представляет собой 0< 0,05 в столбце результата "биномиальный критерий". В таблице 7.2 "+" составляет менее 1,5×10-4 и "-" превышает 1,5×10-4 в столбце результата "наблюдаемая частота ошибок", в то время как "+" представляет собой p > 0,05 и "-" представляет собой p < 0,05 в столбце результата "биномиальный критерий".
Таблица 7.1 Частоты ошибок для некоторых вариантов T7RNAP
Таблица 7.2 Частоты ошибок для некоторых вариантов T7RNAP
[154] Хотя изобретение описано применительно к конкретным вариантам осуществления, могут быть осуществлены различные изменения и эквиваленты могут быть заменены для приспособления к конкретной мутации, материалу, композиции, процессу, стадии или стадиям процесса, тем самым обеспечивая пользу изобретения без отклонения от объема формулы изобретения.
[155] Для всех целей в США каждая публикация и патентный документ, цитированные в настоящем описании, включены в настоящее описание в качестве ссылок, как если бы было конкретно и индивидуально указано, что каждая такая публикация включена в настоящее описание в качестве ссылки. Цитирование публикаций и патентных документов не предназначено для указания на то, что какой-либо из таких документов является соответствующим документом уровня техники, а также не является допущение в отношении его содержания или даты.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> CODEXIS, INC.
ALEXION PHARMACEUTICALS, INC.
<120> ВАРИАНТЫ РНК-ПОЛИМЕРАЗЫ T7
<130> CX8-168USP1
<140> ЕЩЕ НЕ ПРИСВОЕН
<141> ПРИГАЛАЕТСЯ СЮДА
<160> 40
<210> 1
<211> 2649
<212> ДНК
<213> Искусственная последовательность
<220>
<223> T7РНКP дикого типа
<400> 1
atgaacacga ttaacatcgc taagaacgac ttctctgaca tcgaactggc tgctatcccg 60
ttcaacactc tggctgacca ttacggtgag cgtttagctc gcgaacagtt ggcccttgag 120
catgagtctt acgagatggg tgaagcacgc ttccgcaaga tgtttgagcg tcaacttaaa 180
gctggtgagg ttgcggataa cgctgccgcc aagcctctca tcactaccct actccctaag 240
atgattgcac gcatcaacga ctggtttgag gaagtgaaag ctaagcgcgg caagcgcccg 300
acagccttcc agttcctgca agaaatcaag ccggaagccg tagcgtacat caccattaag 360
accactctgg cttgcctaac cagtgctgac aatacaaccg ttcaggctgt agcaagcgca 420
atcggtcggg ccattgagga cgaggctcgc ttcggtcgta tccgtgacct tgaagctaag 480
cacttcaaga aaaacgttga ggaacaactc aacaagcgcg tagggcacgt ctacaagaaa 540
gcatttatgc aagttgtcga ggctgacatg ctctctaagg gtctactcgg tggcgaggcg 600
tggtcttcgt ggcataagga agactctatt catgtaggag tacgctgcat cgagatgctc 660
attgagtcaa ccggaatggt tagcttacac cgccaaaatg ctggcgtagt aggtcaagac 720
tctgagacta tcgaactcgc acctgaatac gctgaggcta tcgcaacccg tgcaggtgcg 780
ctggctggca tctctccgat gttccaacct tgcgtagttc ctcctaagcc gtggactggc 840
attactggtg gtggctattg ggctaacggt cgtcgtcctc tggcgctggt gcgtactcac 900
agtaagaaag cactgatgcg ctacgaagac gtttacatgc ctgaggtgta caaagcgatt 960
aacattgcgc aaaacaccgc atggaaaatc aacaagaaag tcctagcggt cgccaacgta 1020
atcaccaagt ggaagcattg tccggtcgag gacatccctg cgattgagcg tgaagaactc 1080
ccgatgaaac cggaagacat cgacatgaat cctgaggctc tcaccgcgtg gaaacgtgct 1140
gccgctgctg tgtaccgcaa ggacaaggct cgcaagtctc gccgtatcag ccttgagttc 1200
atgcttgagc aagccaataa gtttgctaac cataaggcca tctggttccc ttacaacatg 1260
gactggcgcg gtcgtgttta cgctgtgtca atgttcaacc cgcaaggtaa cgatatgacc 1320
aaaggactgc ttacgctggc gaaaggtaaa ccaatcggta aggaaggtta ctactggctg 1380
aaaatccacg gtgcaaactg tgcgggtgtc gataaggttc cgttccctga gcgcatcaag 1440
ttcattgagg aaaaccacga gaacatcatg gcttgcgcta agtctccact ggagaacact 1500
tggtgggctg agcaagattc tccgttctgc ttccttgcgt tctgctttga gtacgctggg 1560
gtacagcacc acggcctgag ctataactgc tcccttccgc tggcgtttga cgggtcttgc 1620
tctggcatcc agcacttctc cgcgatgctc cgagatgagg taggtggtcg cgcggttaac 1680
ttgcttccta gtgaaaccgt tcaggacatc tacgggattg ttgctaagaa agtcaacgag 1740
attctacaag cagacgcaat caatgggacc gataacgaag tagttaccgt gaccgatgag 1800
aacactggtg aaatctctga gaaagtcaag ctgggcacta aggcactggc tggtcaatgg 1860
ctggcttacg gtgttactcg cagtgtgact aagcgttcag tcatgacgct ggcttacggg 1920
tccaaagagt tcggcttccg tcaacaagtg ctggaagata ccattcagcc agctattgat 1980
tccggcaagg gtctgatgtt cactcagccg aatcaggctg ctggatacat ggctaagctg 2040
atttgggaat ctgtgagcgt gacggtggta gctgcggttg aagcaatgaa ctggcttaag 2100
tctgctgcta agctgctggc tgctgaggtc aaagataaga agactggaga gattcttcgc 2160
aagcgttgcg ctgtgcattg ggtaactcct gatggtttcc ctgtgtggca ggaatacaag 2220
aagcctattc agacgcgctt gaacctgatg ttcctcggtc agttccgctt acagcctacc 2280
attaacacca acaaagatag cgagattgat gcacacaaac aggagtctgg tatcgctcct 2340
aactttgtac acagccaaga cggtagccac cttcgtaaga ctgtagtgtg ggcacacgag 2400
aagtacggaa tcgaatcttt tgcactgatt cacgactcct tcggtaccat tccggctgac 2460
gctgcgaacc tgttcaaagc agtgcgcgaa actatggttg acacatatga gtcttgtgat 2520
gtactggctg atttctacga ccagttcgct gaccagttgc acgagtctca attggacaaa 2580
atgccagcac ttccggctaa aggtaacttg aacctccgtg acatcttaga gtcggacttc 2640
gcgttcgcg 2649
<210> 2
<211> 883
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> T7РНКP дикого типа
<400> 2
Met Asn Thr Ile Asn Ile Ala Lys Asn Asp Phe Ser Asp Ile Glu Leu
1 5 10 15
Ala Ala Ile Pro Phe Asn Thr Leu Ala Asp His Tyr Gly Glu Arg Leu
20 25 30
Ala Arg Glu Gln Leu Ala Leu Glu His Glu Ser Tyr Glu Met Gly Glu
35 40 45
Ala Arg Phe Arg Lys Met Phe Glu Arg Gln Leu Lys Ala Gly Glu Val
50 55 60
Ala Asp Asn Ala Ala Ala Lys Pro Leu Ile Thr Thr Leu Leu Pro Lys
65 70 75 80
Met Ile Ala Arg Ile Asn Asp Trp Phe Glu Glu Val Lys Ala Lys Arg
85 90 95
Gly Lys Arg Pro Thr Ala Phe Gln Phe Leu Gln Glu Ile Lys Pro Glu
100 105 110
Ala Val Ala Tyr Ile Thr Ile Lys Thr Thr Leu Ala Cys Leu Thr Ser
115 120 125
Ala Asp Asn Thr Thr Val Gln Ala Val Ala Ser Ala Ile Gly Arg Ala
130 135 140
Ile Glu Asp Glu Ala Arg Phe Gly Arg Ile Arg Asp Leu Glu Ala Lys
145 150 155 160
His Phe Lys Lys Asn Val Glu Glu Gln Leu Asn Lys Arg Val Gly His
165 170 175
Val Tyr Lys Lys Ala Phe Met Gln Val Val Glu Ala Asp Met Leu Ser
180 185 190
Lys Gly Leu Leu Gly Gly Glu Ala Trp Ser Ser Trp His Lys Glu Asp
195 200 205
Ser Ile His Val Gly Val Arg Cys Ile Glu Met Leu Ile Glu Ser Thr
210 215 220
Gly Met Val Ser Leu His Arg Gln Asn Ala Gly Val Val Gly Gln Asp
225 230 235 240
Ser Glu Thr Ile Glu Leu Ala Pro Glu Tyr Ala Glu Ala Ile Ala Thr
245 250 255
Arg Ala Gly Ala Leu Ala Gly Ile Ser Pro Met Phe Gln Pro Cys Val
260 265 270
Val Pro Pro Lys Pro Trp Thr Gly Ile Thr Gly Gly Gly Tyr Trp Ala
275 280 285
Asn Gly Arg Arg Pro Leu Ala Leu Val Arg Thr His Ser Lys Lys Ala
290 295 300
Leu Met Arg Tyr Glu Asp Val Tyr Met Pro Glu Val Tyr Lys Ala Ile
305 310 315 320
Asn Ile Ala Gln Asn Thr Ala Trp Lys Ile Asn Lys Lys Val Leu Ala
325 330 335
Val Ala Asn Val Ile Thr Lys Trp Lys His Cys Pro Val Glu Asp Ile
340 345 350
Pro Ala Ile Glu Arg Glu Glu Leu Pro Met Lys Pro Glu Asp Ile Asp
355 360 365
Met Asn Pro Glu Ala Leu Thr Ala Trp Lys Arg Ala Ala Ala Ala Val
370 375 380
Tyr Arg Lys Asp Lys Ala Arg Lys Ser Arg Arg Ile Ser Leu Glu Phe
385 390 395 400
Met Leu Glu Gln Ala Asn Lys Phe Ala Asn His Lys Ala Ile Trp Phe
405 410 415
Pro Tyr Asn Met Asp Trp Arg Gly Arg Val Tyr Ala Val Ser Met Phe
420 425 430
Asn Pro Gln Gly Asn Asp Met Thr Lys Gly Leu Leu Thr Leu Ala Lys
435 440 445
Gly Lys Pro Ile Gly Lys Glu Gly Tyr Tyr Trp Leu Lys Ile His Gly
450 455 460
Ala Asn Cys Ala Gly Val Asp Lys Val Pro Phe Pro Glu Arg Ile Lys
465 470 475 480
Phe Ile Glu Glu Asn His Glu Asn Ile Met Ala Cys Ala Lys Ser Pro
485 490 495
Leu Glu Asn Thr Trp Trp Ala Glu Gln Asp Ser Pro Phe Cys Phe Leu
500 505 510
Ala Phe Cys Phe Glu Tyr Ala Gly Val Gln His His Gly Leu Ser Tyr
515 520 525
Asn Cys Ser Leu Pro Leu Ala Phe Asp Gly Ser Cys Ser Gly Ile Gln
530 535 540
His Phe Ser Ala Met Leu Arg Asp Glu Val Gly Gly Arg Ala Val Asn
545 550 555 560
Leu Leu Pro Ser Glu Thr Val Gln Asp Ile Tyr Gly Ile Val Ala Lys
565 570 575
Lys Val Asn Glu Ile Leu Gln Ala Asp Ala Ile Asn Gly Thr Asp Asn
580 585 590
Glu Val Val Thr Val Thr Asp Glu Asn Thr Gly Glu Ile Ser Glu Lys
595 600 605
Val Lys Leu Gly Thr Lys Ala Leu Ala Gly Gln Trp Leu Ala Tyr Gly
610 615 620
Val Thr Arg Ser Val Thr Lys Arg Ser Val Met Thr Leu Ala Tyr Gly
625 630 635 640
Ser Lys Glu Phe Gly Phe Arg Gln Gln Val Leu Glu Asp Thr Ile Gln
645 650 655
Pro Ala Ile Asp Ser Gly Lys Gly Leu Met Phe Thr Gln Pro Asn Gln
660 665 670
Ala Ala Gly Tyr Met Ala Lys Leu Ile Trp Glu Ser Val Ser Val Thr
675 680 685
Val Val Ala Ala Val Glu Ala Met Asn Trp Leu Lys Ser Ala Ala Lys
690 695 700
Leu Leu Ala Ala Glu Val Lys Asp Lys Lys Thr Gly Glu Ile Leu Arg
705 710 715 720
Lys Arg Cys Ala Val His Trp Val Thr Pro Asp Gly Phe Pro Val Trp
725 730 735
Gln Glu Tyr Lys Lys Pro Ile Gln Thr Arg Leu Asn Leu Met Phe Leu
740 745 750
Gly Gln Phe Arg Leu Gln Pro Thr Ile Asn Thr Asn Lys Asp Ser Glu
755 760 765
Ile Asp Ala His Lys Gln Glu Ser Gly Ile Ala Pro Asn Phe Val His
770 775 780
Ser Gln Asp Gly Ser His Leu Arg Lys Thr Val Val Trp Ala His Glu
785 790 795 800
Lys Tyr Gly Ile Glu Ser Phe Ala Leu Ile His Asp Ser Phe Gly Thr
805 810 815
Ile Pro Ala Asp Ala Ala Asn Leu Phe Lys Ala Val Arg Glu Thr Met
820 825 830
Val Asp Thr Tyr Glu Ser Cys Asp Val Leu Ala Asp Phe Tyr Asp Gln
835 840 845
Phe Ala Asp Gln Leu His Glu Ser Gln Leu Asp Lys Met Pro Ala Leu
850 855 860
Pro Ala Lys Gly Asn Leu Asn Leu Arg Asp Ile Leu Glu Ser Asp Phe
865 870 875 880
Ala Phe Ala
<210> 3
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> 6His-T7РНКP
<400> 3
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgctga caatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc tcgcaagtct 1200
cgccgtatca gccttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaagatt ctccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gcagtgtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaagat 1980
accattcagc cagctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 4
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> 6His-T7РНКP
<400> 4
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Asp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Arg Arg Ile Ser Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Asp Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Ser Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Asp Thr Ile Gln Pro Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 5
<211> 3060
<212> ДНК
<213> Искусственная последовательность
<220>
<223> ДНК-матрица
<400> 5
gatcttcttg agatcctttt tttctgcgcg taatctgctg cttgcaaaca aaaaaaccac 60
cgctaccagc ggtggtttgt ttgccggatc aagagctacc aactcttttt ccgaaggtaa 120
ctggcttcag cagagcgcag ataccaaata ctgttcttct agtgtagccg tagttagccc 180
accacttcaa gaactctgta gcaccgccta catacctcgc tctgctaatc ctgttaccag 240
tggctgctgc cagtggcgat aagtcgtgtc ttaccgggtt ggactcaaga cgatagttac 300
cggataaggc gcagcggtcg ggctgaacgg ggggttcgtg cacacagccc agcttggagc 360
gaacgaccta caccgaactg agatacctac agcgtgagct atgagaaagc gccacgcttc 420
ccgaagggag aaaggcggac aggtatccgg taagcggcag ggtcggaaca ggagagcgca 480
cgagggagct tccaggggga aacgcctggt atctttatag tcctgtcggg tttcgccacc 540
tctgacttga gcgtcgattt ttgtgatgct cgtcaggggg gcggagccta tggaaaaacg 600
ccagcaacgc ggccttttta cggttcctgg ccttttgctg gccttttgct cacatgttct 660
ttcctgcgtt atcccctgat tctgtggata accgtattac cgcctttgag tgagctgata 720
ccgctcgccg cagccgaacg accgagcgca gcgagtcagt gagcgaggaa gcggaaggcg 780
agagtaggga actgccaggc atcaaactaa gcagaaggcc cctgacggat ggcctttttg 840
cgtttctaca aactctttct gtgttgtaaa acgacggcca gtcttaagct cgggccccct 900
gggcggttct gataacgagt aatcgttaat ccgcaaataa cgtaaaaacc cgcttcggcg 960
ggttttttta tggggggagt ttagggaaag agcatttgtc agaatattta agggcgcctg 1020
tcactttgct tgatatatga gaattattta accttataaa tgagaaaaaa gcaacgcact 1080
ttaaataaga tacgttgctt tttcgattga tgaacaccta taattaaact attcatctat 1140
tatttatgat tttttgtata tacaatattt ctagtttgtt aaagagaatt aagaaaataa 1200
atctcgaaaa taataaaggg aaaatcagtt tttgatatca aaattataca tgtcaacgat 1260
aatacaaaat ataatacaaa ctataagatg ttatcagtat ttattatgca tttagaataa 1320
attttgtgtc gcccttgtac ttagtcgctg aataatacga ctcactatag cggaacccaa 1380
gctaagcgcc agaactggca ccttcgggtg ccagttgacg aggtggggtt tatcgagatt 1440
tcggcggatg actcccggtt gttcatcaca accgcaagct tttacttaaa tcattaaggt 1500
gacttagtgg acaaaggtga aagtgtgatg aaacccgacc tggacggagg cgcgcccgag 1560
atgagtaggc tgtcccatca ggggaggaat cggggacggc tgaaaggcga gggcgccgaa 1620
gcgagcagag ttcctcccgc tctgcttggc tgggggtgag gggaataccc ttaccactgt 1680
cgcgaaagcg gagagccgtc caggatcccg tcaaaagggc gacaccccat aattagcccg 1740
ggcgaaaggc ccagtctttc gactgagcct ttcgttttat ttgatgcctg gcagttccct 1800
actctcgcat ggggagtccc cacactacca tcggcgctac ggcgtttcac ttctgagttc 1860
ggcatggggt caggtgggac caccgcgcta ctgccgccag gcaaacaagg ggtgttatga 1920
gccatattca ggtataaatg ggctcgcgat aatgttcaga attggttaat tggttgtaac 1980
actgacccct atttgtttat ttttctaaat acattcaaat atgtatccgc tcatgagaca 2040
ataaccctga taaatgcttc aataatattg aaaaaggaag aatatgagta ttcaacattt 2100
ccgtgtcgcc cttattccct tttttgcggc attttgcctt cctgtttttg ctcacccaga 2160
aacgctggtg aaagtaaaag atgctgaaga tcagttgggt gcacgagtgg gttacatcga 2220
actggatctc aacagcggta agatccttga gagttttcgc cccgaagaac gttttccaat 2280
gatgagcact tttaaagttc tgctatgtgg cgcggtatta tcccgtattg acgccgggca 2340
agagcaactc ggtcgccgca tacactattc tcagaatgac ttggttgagt actcaccagt 2400
cacagaaaag catcttacgg atggcatgac agtaagagaa ttatgcagtg ctgccataac 2460
catgagtgat aacactgcgg ccaacttact tctgacaacg atcggaggac cgaaggagct 2520
aaccgctttt ttgcacaaca tgggggatca tgtaactcgc cttgatcgtt gggaaccgga 2580
gctgaatgaa gccataccaa acgacgagcg tgacaccacg atgcctgtag cgatggcaac 2640
aacgttgcgc aaactattaa ctggcgaact acttactcta gcttcccggc aacaattaat 2700
agactggatg gaggcggata aagttgcagg accacttctg cgctcggccc ttccggctgg 2760
ctggtttatt gctgataaat ccggagccgg tgagcgtggt tctcgcggta tcatcgcagc 2820
gctggggcca gatggtaagc cctcccgtat cgtagttatc tacacgacgg ggagtcaggc 2880
aactatggat gaacgaaata gacagatcgc tgagataggt gcctcactga ttaagcattg 2940
gtaaaagcag agcattacgc tgacttgacg ggacggcgca agctcatgac caaaatccct 3000
taacgtgagt tacgcgcgcg tcgttccact gagcgtcaga ccccgtagaa aagatcaaag 3060
<210> 6
<211> 334
<212> РНК
<213> Искусственная последовательность
<220>
<223> РНК-транскрипт
<400> 6
gcggaaccca agcuaagcgc cagaacuggc accuucgggu gccaguugac gagguggggu 60
uuaucgagau uucggcggau gacucccggu uguucaucac aaccgcaagc uuuuacuuaa 120
aucauuaagg ugacuuagug gacaaaggug aaagugugau gaaacccgac cuggacggag 180
gcgcgcccga gaugaguagg cugucccauc aggggaggaa ucggggacgg cugaaaggcg 240
agggcgccga agcgagcaga guuccucccg cucugcuugg cuggggguga ggggaauacc 300
cuuaccacug ucgcgaaagc ggagagccgu ccag 334
<210> 7
<211> 16
<212> РНК
<213> Искусственная последовательность
<220>
<223> Продукт расщепления РНК
<400> 7
gcggaaccca agcuaa 16
<210> 8
<211> 4671
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Субстрат и т.д.
<400> 8
gagttacgcg cgcgtcgttc cactgagcgt cagaccccgt agaaaagatc aaaggatctt 60
cttgagatcc tttttttctg cgcgtaatct gctgcttgca aacaaaaaaa ccaccgctac 120
cagcggtggt ttgtttgccg gatcaagagc taccaactct ttttccgaag gtaactggct 180
tcagcagagc gcagatacca aatactgttc ttctagtgta gccgtagtta gcccaccact 240
tcaagaactc tgtagcaccg cctacatacc tcgctctgct aatcctgtta ccagtggctg 300
ctgccagtgg cgataagtcg tgtcttaccg ggttggactc aagacgatag ttaccggata 360
aggcgcagcg gtcgggctga acggggggtt cgtgcacaca gcccagcttg gagcgaacga 420
cctacaccga actgagatac ctacagcgtg agctatgaga aagcgccacg cttcccgaag 480
ggagaaaggc ggacaggtat ccggtaagcg gcagggtcgg aacaggagag cgcacgaggg 540
agcttccagg gggaaacgcc tggtatcttt atagtcctgt cgggtttcgc cacctctgac 600
ttgagcgtcg atttttgtga tgctcgtcag gggggcggag cctatggaaa aacgccagca 660
acgcggcctt tttacggttc ctggcctttt gctggccttt tgctcacatg ttctttcctg 720
cgttatcccc tgattctgtg gataaccgta ttaccgcctt tgagtgagct gataccgctc 780
gccgcagccg aacgaccgag cgcagcgagt cagtgagcga ggaagcggaa ggcgagagta 840
gggaactgcc aggcatcaaa ctaagcagaa ggcccctgac ggatggcctt tttgcgtttc 900
tacaaactct ttctgtgttg taaaacgacg gccagtctta agctcgggcc ccctgggcgg 960
ttctgataac gagtaatcgt taatccgcaa ataacgtaaa aacccgcttc ggcgggtttt 1020
tttatggggg gagtttaggg aaagagcatt tgtcagaata tttaagggcg cctgtcactt 1080
tgcttgatat atgagaatta tttaacctta taaatgagaa aaaagcaacg cactttaaat 1140
aagatacgtt gctttttcga ttgatgaaca cctataatta aactattcat ctattattta 1200
tgattttttg tatatacaat atttctagtt tgttaaagag aattaagaaa ataaatctcg 1260
aaaataataa agggaaaatc agtttttgat atcaaaatta tacatgtcaa cgataataca 1320
aaatataata caaactataa gatgttatca gtatttatta tgcatttaga atacgtactc 1380
agcgtctggg ttccccatcg gtgatgtcgt ataagagacg tataggagac ctatagtgtc 1440
ttcggggtaa tacgactcac tatagcggaa cccaagcttg gcattccggt actgttggta 1500
aagccaccat ggaagatgcg aagaacataa agaaaggtcc cgccccattt tacccactcg 1560
aggatggaac agctggggag caactgcaca aggccatgaa gcgctatgcg ttggtgccgg 1620
gaaccatcgc gttcaccgac gcacacatcg aagtgaacat cacttacgcc gagtactttg 1680
agatgagcgt caggctggcc gaggctatga agcgatacgg tctgaacacc aaccaccgga 1740
tcgtggtctg ctctgaaaac agcctgcagt tcttcatgcc ggtcctgggg gccctgttca 1800
tcggcgtggc cgtggcaccc gccaacgata tctacaacga gagagaattg ctgaactcga 1860
tgaacatctc ccagcctacc gtggtgttcg tgtcgaagaa ggggttgcag aagatcctga 1920
acgtgcagaa gaagctgccc atcattcaaa agattatcat tatggattcc aaaaccgact 1980
accagggttt ccagtcaatg tataccttcg tgacctccca tctgccccct ggcttcaacg 2040
aatacgactt cgtgcctgaa agcttcgacc gcgacaagac gatcgccctc atcatgaact 2100
cgtccggctc gaccgggctg cccaaaggag tggccctgcc acaccggacc gcttgcgtgc 2160
ggttctccca cgcccgggac cctattttcg gcaatcagat cattccggac actgccatcc 2220
tgagcgtggt ccccttccat cacgggtttg ggatgtttac cactctgggc tacctcatct 2280
gcggattcag ggtggtgctg atgtaccggt tcgaggaaga acttttcctg cggagcctgc 2340
aggattacaa gatccagtcc gccctcctcg tgccaaccct cttctcattc ttcgctaagt 2400
ccactctcat cgataagtac gacctgtcga atctccacga aattgcgtcc ggtggtgcac 2460
cgctgtccaa ggaggtcggc gaagccgtgg ccaagcgctt ccacctcccg ggaatacgcc 2520
agggatacgg cctgactgaa acgaccagcg cgattctgat caccccggag ggcgacgaca 2580
agccgggtgc cgtggggaaa gtggtgccgt tcttcgaagc aaaggtcgtg gatctggata 2640
ccggaaagac tctgggcgtg aaccagagag gggaactttg tgtgcgcgga ccgatgatta 2700
tgtccggata tgtcaacaac cccgaggcca ctaatgccct gatcgacaag gacggatggt 2760
tgcatagcgg cgacatcgca tactgggacg aggacgagca ctttttcatt gtggatcggc 2820
tcaagtccct gatcaagtac aagggatacc aggtcgcccc tgccgaactt gagtccatcc 2880
tgctgcaaca tccgaacatt ttcgacgcgg gcgtcgctgg ccttcctgat gatgacgccg 2940
gagagctgcc cgcggccgtg gtggtgctcg aacacggaaa aactatgacc gagaaggaaa 3000
tcgtggacta cgtggcgtca caagtcacca ctgccaagaa actgcgcggc ggagtcgtgt 3060
tcgtggacga ggtgcccaag ggcctgaccg gaaagctgga cgctagaaag atccgggaga 3120
tcctgattaa ggccaagaag ggaggaaagt ccaagctctg agatctagag ggccctattc 3180
tatagtgtca cctaaatgct aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3240
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3300
gaattcaaaa gaagaccata tacgtctcct ataggtctcg tatactgctt cctatacgac 3360
atcaccgatg gggaacacga agcgatccag ccccccttat tagcccgggc gaaaggccca 3420
gtctttcgac tgagcctttc gttttatttg atgcctggca gttccctact ctcgcatggg 3480
gagtccccac actaccatcg gcgctacggc gtttcacttc tgagttcggc atggggtcag 3540
gtgggaccac cgcgctactg ccgccaggca aacaaggggt gttatgagcc atattcaggt 3600
ataaatgggc tcgcgataat gttcagaatt ggttaattgg ttgtaacact gacccctatt 3660
tgtttatttt tctaaataca ttcaaatatg tatccgctca tgagacaata accctgataa 3720
atgcttcaat aatattgaaa aaggaagaat atgagtattc aacatttccg tgtcgccctt 3780
attccctttt ttgcggcatt ttgccttcct gtttttgctc acccagaaac gctggtgaaa 3840
gtaaaagatg ctgaagatca gttgggtgca cgagtgggtt acatcgaact ggatctcaac 3900
agcggtaaga tccttgagag ttttcgcccc gaagaacgtt ttccaatgat gagcactttt 3960
aaagttctgc tatgtggcgc ggtattatcc cgtattgacg ccgggcaaga gcaactcggt 4020
cgccgcatac actattctca gaatgacttg gttgagtact caccagtcac agaaaagcat 4080
cttacggatg gcatgacagt aagagaatta tgcagtgctg ccataaccat gagtgataac 4140
actgcggcca acttacttct gacaacgatc ggaggaccga aggagctaac cgcttttttg 4200
cacaacatgg gggatcatgt aactcgcctt gatcgttggg aaccggagct gaatgaagcc 4260
ataccaaacg acgagcgtga caccacgatg cctgtagcga tggcaacaac gttgcgcaaa 4320
ctattaactg gcgaactact tactctagct tcccggcaac aattaataga ctggatggag 4380
gcggataaag ttgcaggacc acttctgcgc tcggcccttc cggctggctg gtttattgct 4440
gataaatccg gagccggtga gcgtggttct cgcggtatca tcgcagcgct ggggccagat 4500
ggtaagccct cccgtatcgt agttatctac acgacgggga gtcaggcaac tatggatgaa 4560
cgaaatagac agatcgctga gataggtgcc tcactgatta agcattggta agcagagcat 4620
tacgctgact tgacgggacg gcgcaagctc atgaccaaaa tcccttaacg t 4671
<210> 9
<211> 1697
<212> РНК
<213> Искусственная последовательность
<220>
<223> Субстрат и т.д.
<400> 9
gcggaaccca agcuuggcau uccgguacug uugguaaagc caccauggaa gaugcgaaga 60
acauaaagaa aggucccgcc ccauuuuacc cacucgagga uggaacagcu ggggagcaac 120
ugcacaaggc caugaagcgc uaugcguugg ugccgggaac caucgcguuc accgacgcac 180
acaucgaagu gaacaucacu uacgccgagu acuuugagau gagcgucagg cuggccgagg 240
cuaugaagcg auacggucug aacaccaacc accggaucgu ggucugcucu gaaaacagcc 300
ugcaguucuu caugccgguc cugggggccc uguucaucgg cguggccgug gcacccgcca 360
acgauaucua caacgagaga gaauugcuga acucgaugaa caucucccag ccuaccgugg 420
uguucguguc gaagaagggg uugcagaaga uccugaacgu gcagaagaag cugcccauca 480
uucaaaagau uaucauuaug gauuccaaaa ccgacuacca ggguuuccag ucaauguaua 540
ccuucgugac cucccaucug cccccuggcu ucaacgaaua cgacuucgug ccugaaagcu 600
ucgaccgcga caagacgauc gcccucauca ugaacucguc cggcucgacc gggcugccca 660
aaggaguggc ccugccacac cggaccgcuu gcgugcgguu cucccacgcc cgggacccua 720
uuuucggcaa ucagaucauu ccggacacug ccauccugag cguggucccc uuccaucacg 780
gguuugggau guuuaccacu cugggcuacc ucaucugcgg auucagggug gugcugaugu 840
accgguucga ggaagaacuu uuccugcgga gccugcagga uuacaagauc caguccgccc 900
uccucgugcc aacccucuuc ucauucuucg cuaaguccac ucucaucgau aaguacgacc 960
ugucgaaucu ccacgaaauu gcguccggug gugcaccgcu guccaaggag gucggcgaag 1020
ccguggccaa gcgcuuccac cucccgggaa uacgccaggg auacggccug acugaaacga 1080
ccagcgcgau ucugaucacc ccggagggcg acgacaagcc gggugccgug gggaaagugg 1140
ugccguucuu cgaagcaaag gucguggauc uggauaccgg aaagacucug ggcgugaacc 1200
agagagggga acuuugugug cgcggaccga ugauuauguc cggauauguc aacaaccccg 1260
aggccacuaa ugcccugauc gacaaggacg gaugguugca uagcggcgac aucgcauacu 1320
gggacgagga cgagcacuuu uucauugugg aucggcucaa gucccugauc aaguacaagg 1380
gauaccaggu cgccccugcc gaacuugagu ccauccugcu gcaacauccg aacauuuucg 1440
acgcgggcgu cgcuggccuu ccugaugaug acgccggaga gcugcccgcg gccguggugg 1500
ugcucgaaca cggaaaaacu augaccgaga aggaaaucgu ggacuacgug gcgucacaag 1560
ucaccacugc caagaaacug cgcggcggag ucguguucgu ggacgaggug cccaagggcc 1620
ugaccggaaa gcuggacgcu agaaagaucc gggagauccu gauuaaggcc aagaagggag 1680
gaaaguccaa gcucuga 1697
<210> 10
<211> 4945
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Субстрат и т.д.
<400> 10
ttaccaatgc ttaatcagtg aggcacctat ctcagcgatc tgtctatttc gttcatccat 60
agttgcctga ctccccgtcg tgtagataac tacgatacgg gagggcttac catctggccc 120
cagcgctgcg atgataccgc gagaaccacg ctcaccggct ccggatttat cagcaataaa 180
ccagccagcc ggaagggccg agcgcagaag tggtcctgca actttatccg cctccatcca 240
gtctattaat tgttgccggg aagctagagt aagtagttcg ccagttaata gtttgcgcaa 300
cgttgttgcc atcgctacag gcatcgtggt gtcacgctcg tcgtttggta tggcttcatt 360
cagctccggt tcccaacgat caaggcgagt tacatgatcc cccatgttgt gcaaaaaagc 420
ggttagctcc ttcggtcctc cgatcgttgt cagaagtaag ttggccgcag tgttatcact 480
catggttatg gcagcactgc ataattctct tactgtcatg ccatccgtaa gatgcttttc 540
tgtgactggt gagtactcaa ccaagtcatt ctgagaatag tgtatgcggc gaccgagttg 600
ctcttgcccg gcgtcaatac gggataatac cgcgccacat agcagaactt taaaagtgct 660
catcattgga aaacgttctt cggggcgaaa actctcaagg atcttaccgc tgttgagatc 720
cagttcgatg taacccactc gtgcacccaa ctgatcttca gcatctttta ctttcaccag 780
cgtttctggg tgagcaaaaa caggaaggca aaatgccgca aaaaagggaa taagggcgac 840
acggaaatgt tgaatactca tattcttcct ttttcaatat tattgaagca tttatcaggg 900
ttattgtctc atgagcggat acatatttga atgtatttag aaaaataaac aaataggggt 960
cagtgttaca accaattaac caattctgaa cattatcgcg agcccattta tacctgaata 1020
tggctcataa caccccttgt ttgcctggcg gcagtagcgc ggtggtccca cctgacccca 1080
tgccgaactc agaagtgaaa cgccgtagcg ccgatggtag tgtggggact ccccatgcga 1140
gagtagggaa ctgccaggca tcaaataaaa cgaaaggctc agtcgaaaga ctgggccttt 1200
cgcccgggct aattatgggg tgtcgccctt cgctgaaggg gtaatacgac tcactatagg 1260
gaaataagag agaaaagaag agtaagaaga aatataagag ccaccatgga agatgcgaag 1320
aacataaaga aaggtcccgc cccattttac ccactcgagg atggaacagc tggggagcaa 1380
ctgcacaagg ccatgaagcg ctatgcgttg gtgccgggaa ccatcgcgtt caccgacgca 1440
cacatcgaag tgaacatcac ttacgccgag tactttgaga tgagcgtcag gctggccgag 1500
gctatgaagc gatacggtct gaacaccaac caccggatcg tggtctgctc tgaaaacagc 1560
ctgcagttct tcatgccggt cctgggggcc ctgttcatcg gcgtggccgt ggcacccgcc 1620
aacgatatct acaacgagag agaattgctg aactcgatga acatctccca gcctaccgtg 1680
gtgttcgtgt cgaagaaggg gttgcagaag atcctgaacg tgcagaagaa gctgcccatc 1740
attcaaaaga ttatcattat ggattccaaa accgactacc agggtttcca gtcaatgtat 1800
accttcgtga cctcccatct gccccctggc ttcaacgaat acgacttcgt gcctgaaagc 1860
ttcgaccgcg acaagacgat cgccctcatc atgaactcgt ccggctcgac cgggctgccc 1920
aaaggagtgg ccctgccaca ccggaccgct tgcgtgcggt tctcccacgc ccgggaccct 1980
attttcggca atcagatcat tccggacact gccatcctga gcgtggtccc cttccatcac 2040
gggtttggga tgtttaccac tctgggctac ctcatctgcg gattcagggt ggtgctgatg 2100
taccggttcg aggaagaact tttcctgcgg agcctgcagg attacaagat ccagtccgcc 2160
ctcctcgtgc caaccctctt ctcattcttc gctaagtcca ctctcatcga taagtacgac 2220
ctgtcgaatc tccacgaaat tgcgtccggt ggtgcaccgc tgtccaagga ggtcggcgaa 2280
gccgtggcca agcgcttcca cctcccggga attcgccagg gatacggcct gactgaaacg 2340
accagcgcga ttctgatcac cccggagggc gacgacaagc cgggtgccgt ggggaaagtg 2400
gtgccgttct tcgaagcaaa ggtcgtggat ctggataccg gaaagactct gggcgtgaac 2460
cagagagggg aactttgtgt gcgcggaccg atgattatgt ccggatatgt caacaacccc 2520
gaggccacta atgccctgat cgacaaggac ggatggttgc atagcggcga catcgcatac 2580
tgggacgagg acgagcactt tttcattgtg gatcggctca agtccctgat caagtacaag 2640
ggataccagg tcgcccctgc cgaacttgag tccatcctgc tgcaacatcc gaacattttc 2700
gacgcgggcg tcgctggcct tcctgatgat gacgccggag agctgcccgc ggccgtggtg 2760
gtgctcgaac acggaaaaac tatgaccgag aaggaaatcg tggactacgt ggcgtcacaa 2820
gtcaccactg ccaagaaact gcgcggcgga gtcgtgttcg tggacgaggt gcccaagggc 2880
ctgaccggaa agctggacgc tagaaagatc cgggagatcc tgattaaggc caagaaggga 2940
ggaaagtcca agctctgaat ctgctgcctt ctgcggggct tgccttctgg ccatgccctt 3000
cttctctccc ttgcacctgt acctcttggt ctttgaataa agcctgagta ggaagtgagg 3060
gaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 3120
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaagga agagcgctgc 3180
ctccttcaga gtctgctcac gagctcggtc tcggatcccc tatacgacat caccgatggg 3240
gaacacgaag cgatccagcc ccccttatta gcccgggcga aaggcccagt ctttcgactg 3300
agcctttcgt tttatttgat gcctggcagt tccctactct cgcatgggga gtccccacac 3360
taccatcggc gctacggcgt ttcacttctg agttcggcat ggggtcaggt gggaccaccg 3420
cgctactgcc gccaggcaaa caaggggtgt tatgagccat attcaggtat aaatgggctc 3480
gcgaaaacgt caaaagggcg acacaaaatt tattctaaat gcataataaa tactgataac 3540
atcttatagt ttgtattata ttttgtatta tcgttgacat gtataatttt gatatcaaaa 3600
actgattttc cctttattat tttcgagatt tattttctta attctcttta acaaactaga 3660
aatattgtat atacaaaaaa tcataaataa tagatgaata gtttaattat aggtgttcat 3720
caatcgaaaa agcaacgtat cttatttaaa gtgcgttgct tttttctcat ttataaggtt 3780
aaataattct catatatcaa gcaaagtgac aggcgccctt aaatattctg acaaatgctc 3840
tttccctaaa ctccccccat aaaaaaaccc gccgaagcgg gtttttacgt tatttgcgga 3900
ttaacgatta ctcgttatca gaaccgccca gggggcccga gcttaagact ggccgtcgtt 3960
ttacaacaca gaaagagttt gtagaaacgc aaaaaggcca tccgtcaggg gccttctgct 4020
tagtttgatg cctggcagtt ccctactctc gccttccgct tcctcgctca ctgactcgct 4080
gcgctcggtc gttcggctgc ggcgagcggt atcagctcac tcaaaggcgg taatacggtt 4140
atccacagaa tcaggggata acgcaggaaa gaacatgtga gcaaaaggcc agcaaaaggc 4200
caggaaccgt aaaaaggccg cgttgctggc gtttttccat aggctccgcc cccctgacga 4260
gcatcacaaa aatcgacgct caagtcagag gtggcgaaac ccgacaggac tataaagata 4320
ccaggcgttt ccccctggaa gctccctcgt gcgctctcct gttccgaccc tgccgcttac 4380
cggatacctg tccgcctttc tcccttcggg aagcgtggcg ctttctcata gctcacgctg 4440
taggtatctc agttcggtgt aggtcgttcg ctccaagctg ggctgtgtgc acgaaccccc 4500
cgttcagccc gaccgctgcg ccttatccgg taactatcgt cttgagtcca acccggtaag 4560
acacgactta tcgccactgg cagcagccac tggtaacagg attagcagag cgaggtatgt 4620
aggcggtgct acagagttct tgaagtggtg ggctaactac ggctacacta gaagaacagt 4680
atttggtatc tgcgctctgc tgaagccagt taccttcgga aaaagagttg gtagctcttg 4740
atccggcaaa caaaccaccg ctggtagcgg tggttttttt gtttgcaagc agcagattac 4800
gcgcagaaaa aaaggatctc aagaagatcc tttgatcttt tctacggggt ctgacgctca 4860
gtggaacgac gcgcgcgtaa ctcacgttaa gggattttgg tcatgagctt gcgccgtccc 4920
gtcaagtcag cgtaatgctc tgctt 4945
<210> 11
<211> 1955
<212> РНК
<213> Искусственная последовательность
<220>
<223> Субстрат и т.д.
<400> 11
gggaaauaag agagaaaaga agaguaagaa gaaauauaag agccaccaug gaagaugcga 60
agaacauaaa gaaagguccc gccccauuuu acccacucga ggauggaaca gcuggggagc 120
aacugcacaa ggccaugaag cgcuaugcgu uggugccggg aaccaucgcg uucaccgacg 180
cacacaucga agugaacauc acuuacgccg aguacuuuga gaugagcguc aggcuggccg 240
aggcuaugaa gcgauacggu cugaacacca accaccggau cguggucugc ucugaaaaca 300
gccugcaguu cuucaugccg guccuggggg cccuguucau cggcguggcc guggcacccg 360
ccaacgauau cuacaacgag agagaauugc ugaacucgau gaacaucucc cagccuaccg 420
ugguguucgu gucgaagaag ggguugcaga agauccugaa cgugcagaag aagcugccca 480
ucauucaaaa gauuaucauu auggauucca aaaccgacua ccaggguuuc cagucaaugu 540
auaccuucgu gaccucccau cugcccccug gcuucaacga auacgacuuc gugccugaaa 600
gcuucgaccg cgacaagacg aucgcccuca ucaugaacuc guccggcucg accgggcugc 660
ccaaaggagu ggcccugcca caccggaccg cuugcgugcg guucucccac gcccgggacc 720
cuauuuucgg caaucagauc auuccggaca cugccauccu gagcgugguc cccuuccauc 780
acggguuugg gauguuuacc acucugggcu accucaucug cggauucagg guggugcuga 840
uguaccgguu cgaggaagaa cuuuuccugc ggagccugca ggauuacaag auccaguccg 900
cccuccucgu gccaacccuc uucucauucu ucgcuaaguc cacucucauc gauaaguacg 960
accugucgaa ucuccacgaa auugcguccg guggugcacc gcuguccaag gaggucggcg 1020
aagccguggc caagcgcuuc caccucccgg gaauucgcca gggauacggc cugacugaaa 1080
cgaccagcgc gauucugauc accccggagg gcgacgacaa gccgggugcc guggggaaag 1140
uggugccguu cuucgaagca aaggucgugg aucuggauac cggaaagacu cugggcguga 1200
accagagagg ggaacuuugu gugcgcggac cgaugauuau guccggauau gucaacaacc 1260
ccgaggccac uaaugcccug aucgacaagg acggaugguu gcauagcggc gacaucgcau 1320
acugggacga ggacgagcac uuuuucauug uggaucggcu caagucccug aucaaguaca 1380
agggauacca ggucgccccu gccgaacuug aguccauccu gcugcaacau ccgaacauuu 1440
ucgacgcggg cgucgcuggc cuuccugaug augacgccgg agagcugccc gcggccgugg 1500
uggugcucga acacggaaaa acuaugaccg agaaggaaau cguggacuac guggcgucac 1560
aagucaccac ugccaagaaa cugcgcggcg gagucguguu cguggacgag gugcccaagg 1620
gccugaccgg aaagcuggac gcuagaaaga uccgggagau ccugauuaag gccaagaagg 1680
gaggaaaguc caagcucuga aucugcugcc uucugcgggg cuugccuucu ggccaugccc 1740
uucuucucuc ccuugcaccu guaccucuug gucuuugaau aaagccugag uaggaaguga 1800
gggaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa 1860
aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaaaag gaagagcgcu 1920
gccuccuuca gagucugcuc acgagcucgg ucucg 1955
<210> 12
<211> 57
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Субстрат и т.д.
<400> 12
tctagaggcc agcctggcca taaggagata tacatcggta ctgttggtaa agccacc 57
<210> 13
<211> 50
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Субстрат и т.д.
<400> 13
taatgaggcc aaactggcca ccatcaccat cagagcttgg actttcctcc 50
<210> 14
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 14
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgctga caatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc tcgcaagtct 1200
cgccgtatca gccttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaagatt ctccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gcagtgtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaagat 1980
accattcagt gggctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 15
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 15
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Asp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Arg Arg Ile Ser Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Asp Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Ser Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Asp Thr Ile Gln Trp Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 16
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 16
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgctga taatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc tcgcaagtct 1200
agacgtatca gccttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaagatt ctccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gctgggtgac aaagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaaacc 1980
accattcagt gggctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 17
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 17
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Asp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Arg Arg Ile Ser Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Asp Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Trp Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Thr Thr Ile Gln Trp Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 18
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 18
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgctga taatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc tcgcaagtct 1200
cgccgtatca gccttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaattgt ctccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gcagtgtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaatgg 1980
accattcagt gggctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 19
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 19
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Asp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Arg Arg Ile Ser Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Leu Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Ser Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Trp Thr Ile Gln Trp Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 20
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 20
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgcttg gaatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc tcgcaagtct 1200
agtcgtatca gccttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaacggt ctccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gcagtgtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaagat 1980
accattcagt gggctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 21
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 21
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Trp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Ser Arg Ile Ser Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Arg Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Ser Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Asp Thr Ile Gln Trp Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 22
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 22
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgcttg gaatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc tcgcaagtct 1200
agacgtatca gccttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaatggt ctccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gcagtgtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaagat 1980
accattcagt gggctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 23
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 23
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Trp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Arg Arg Ile Ser Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Trp Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Ser Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Asp Thr Ile Gln Trp Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 24
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 24
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgcttg gaatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc tcgcaagtct 1200
agtcgtatca gccttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaatggt ctccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gcagtgtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaagat 1980
accattcagt gggctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 25
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 25
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Trp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Ser Arg Ile Ser Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Trp Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Ser Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Asp Thr Ile Gln Trp Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 26
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 26
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgcttg gaatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc tcgcaagtct 1200
atacgtatca gccttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaagatt ctccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gcagtgtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaagat 1980
accattcagt gggctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 27
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 27
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Trp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Ile Arg Ile Ser Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Asp Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Ser Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Asp Thr Ile Gln Trp Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 28
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 28
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtataga caatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga agacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca gagacaaggc tcgcaagtct 1200
cgccgtatct atcttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgattggac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaagatt ccccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gcagtgtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaagat 1980
accattcagt gggctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 29
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 29
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ile Asp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Arg Asp Lys Ala Arg Lys Ser
385 390 395 400
Arg Arg Ile Tyr Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Trp Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Asp Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Ser Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Asp Thr Ile Gln Trp Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 30
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 30
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgctga caatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc gcgcaagtct 1200
cgccgtatca gccttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaatggt ctccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gctgggtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaattc 1980
accattcagc ctgctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 31
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 31
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Asp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Arg Arg Ile Ser Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Trp Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Trp Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Phe Thr Ile Gln Pro Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 32
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 32
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgctga caatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc gcgcaagtct 1200
cgccgtatca gccttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaatatt ctccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gctgggtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaatac 1980
accattcagc ctgctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 33
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 33
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Asp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Arg Arg Ile Ser Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Tyr Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Trp Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Tyr Thr Ile Gln Pro Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 34
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 34
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgcgga caatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc tcgcaagtct 1200
gttcgtatct atcttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaagatc tcccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gcagtgtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaagac 1980
accattcagt gggctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 35
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 35
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Asp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Val Arg Ile Tyr Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Asp Leu Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Ser Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Asp Thr Ile Gln Trp Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 36
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 36
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgcgga caatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtccg ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca gagacaaggc tcgcaagtct 1200
gttcgtatct atcttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaagatc tcccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gcagtgtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaagat 1980
actattcagt gggctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 37
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 37
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Asp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Arg Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Arg Asp Lys Ala Arg Lys Ser
385 390 395 400
Val Arg Ile Tyr Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Asp Leu Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Ser Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Asp Thr Ile Gln Trp Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 38
<211> 2673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 38
atgcaccatc accatcacca tatgaacacg attaacatcg ctaagaacga cttctctgac 60
atcgaactgg ctgctatccc gttcaacact ctggctgacc attacggtga gcgtttagct 120
cgcgaacagt tggcccttga gcatgagtct tacgagatgg gtgaagcacg cttccgcaag 180
atgtttgagc gtcaacttaa agctggtgag gttgcggata acgctgccgc caagcctctc 240
atcactaccc tactccctaa gatgattgca cgcatcaacg actggtttga ggaagtgaaa 300
gctaagcgcg gcaagcgccc gacagccttc cagttcctgc aagaaatcaa gccggaagcc 360
gtagcgtaca tcaccattaa gaccactctg gcttgcctaa ccagtgctga caatacaacc 420
gttcaggctg tagcaagcgc aatcggtcgg gccattgagg acgaggctcg cttcggtcgt 480
atccgtgacc ttgaagctaa gcacttcaag aaaaacgttg aggaacaact caacaagcgc 540
gtagggcacg tctacaagaa agcatttatg caagttgtcg aggctgacat gctctctaag 600
ggtctactcg gtggcgaggc gtggtcttcg tggcataagg aagactctat tcatgtagga 660
gtacgctgca tcgagatgct cattgagtca accggaatgg ttagcttaca ccgccaaaat 720
gctggcgtag taggtcaaga ctctgagact atcgaactcg cacctgaata cgctgaggct 780
atcgcaaccc gtgcaggtgc gctggctggc atctctccga tgttccaacc ttgcgtagtt 840
cctcctaagc cgtggactgg cattactggt ggtggctatt gggctaacgg tcgtcgtcct 900
ctggcgctgg tgcgtactca cagtaagaaa gcactgatgc gctacgaaga cgtttacatg 960
cctgaggtgt acaaagcgat taacattgcg caaaacaccg catggaaaat caacaagaaa 1020
gtcctagcgg tcgccaacgt aatcaccaag tggaagcatt gtccggtcga ggacatccct 1080
gcgattgagc gtgaagaact cccgatgaaa ccggaagaca tcgacatgaa tcctgaggct 1140
ctcaccgcgt ggaaacgtgc tgccgctgct gtgtaccgca aggacaaggc gcgcaagtct 1200
cgccgtatca gccttgagtt catgcttgag caagccaata agtttgctaa ccataaggcc 1260
atctggttcc cttacaacat ggactggcgc ggtcgtgttt acgctgtgtc aatgttcaac 1320
ccgcaaggta acgatatgac caaaggactg cttacgctgg cgaaaggtaa accaatcggt 1380
aaggaaggtt actactggct gaaaatccac ggtgcaaact gtgcgggtgt cgataaggtt 1440
ccgttccctg agcgcatcaa gttcattgag gaaaaccacg agaacatcat ggcttgcgct 1500
aagtctccac tggagaacac ttggtgggct gagcaatatt ctccgttctg cttccttgcg 1560
ttctgctttg agtacgctgg ggtacagcac cacggcctga gctataactg ctcccttccg 1620
ctggcgtttg acgggtcttg ctctggcatc cagcacttct ccgcgatgct ccgagatgag 1680
gtaggtggtc gcgcggttaa cttgcttcct agtgaaaccg ttcaggacat ctacgggatt 1740
gttgctaaga aagtcaacga gattctacaa gcagacgcaa tcaatgggac cgataacgaa 1800
gtagttaccg tgaccgatga gaacactggt gaaatctctg agaaagtcaa gctgggcact 1860
aaggcactgg ctggtcaatg gctggcttac ggtgttactc gctgggtgac taagcgttca 1920
gtcatgacgc tggcttacgg gtccaaagag ttcggcttcc gtcaacaagt gctggaattc 1980
accattcagc ctgctattga ttccggcaag ggtctgatgt tcactcagcc gaatcaggct 2040
gctggataca tggctaagct gatttgggaa tctgtgagcg tgacggtggt agctgcggtt 2100
gaagcaatga actggcttaa gtctgctgct aagctgctgg ctgctgaggt caaagataag 2160
aagactggag agattcttcg caagcgttgc gctgtgcatt gggtaactcc tgatggtttc 2220
cctgtgtggc aggaatacaa gaagcctatt cagacgcgct tgaacctgat gttcctcggt 2280
cagttccgct tacagcctac cattaacacc aacaaagata gcgagattga tgcacacaaa 2340
caggagtctg gtatcgctcc taactttgta cacagccaag acggtagcca ccttcgtaag 2400
actgtagtgt gggcacacga gaagtacgga atcgaatctt ttgcactgat tcacgactcc 2460
ttcggtacca ttccggctga cgctgcgaac ctgttcaaag cagtgcgcga aactatggtt 2520
gacacatatg agtcttgtga tgtactggct gatttctacg accagttcgc tgaccagttg 2580
cacgagtctc aattggacaa aatgccagca cttccggcta aaggtaactt gaacctccgt 2640
gacatcttag agtcggactt cgcgttcgcg taa 2673
<210> 39
<211> 890
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Вариант T7РНКP
<400> 39
Met His His His His His His Met Asn Thr Ile Asn Ile Ala Lys Asn
1 5 10 15
Asp Phe Ser Asp Ile Glu Leu Ala Ala Ile Pro Phe Asn Thr Leu Ala
20 25 30
Asp His Tyr Gly Glu Arg Leu Ala Arg Glu Gln Leu Ala Leu Glu His
35 40 45
Glu Ser Tyr Glu Met Gly Glu Ala Arg Phe Arg Lys Met Phe Glu Arg
50 55 60
Gln Leu Lys Ala Gly Glu Val Ala Asp Asn Ala Ala Ala Lys Pro Leu
65 70 75 80
Ile Thr Thr Leu Leu Pro Lys Met Ile Ala Arg Ile Asn Asp Trp Phe
85 90 95
Glu Glu Val Lys Ala Lys Arg Gly Lys Arg Pro Thr Ala Phe Gln Phe
100 105 110
Leu Gln Glu Ile Lys Pro Glu Ala Val Ala Tyr Ile Thr Ile Lys Thr
115 120 125
Thr Leu Ala Cys Leu Thr Ser Ala Asp Asn Thr Thr Val Gln Ala Val
130 135 140
Ala Ser Ala Ile Gly Arg Ala Ile Glu Asp Glu Ala Arg Phe Gly Arg
145 150 155 160
Ile Arg Asp Leu Glu Ala Lys His Phe Lys Lys Asn Val Glu Glu Gln
165 170 175
Leu Asn Lys Arg Val Gly His Val Tyr Lys Lys Ala Phe Met Gln Val
180 185 190
Val Glu Ala Asp Met Leu Ser Lys Gly Leu Leu Gly Gly Glu Ala Trp
195 200 205
Ser Ser Trp His Lys Glu Asp Ser Ile His Val Gly Val Arg Cys Ile
210 215 220
Glu Met Leu Ile Glu Ser Thr Gly Met Val Ser Leu His Arg Gln Asn
225 230 235 240
Ala Gly Val Val Gly Gln Asp Ser Glu Thr Ile Glu Leu Ala Pro Glu
245 250 255
Tyr Ala Glu Ala Ile Ala Thr Arg Ala Gly Ala Leu Ala Gly Ile Ser
260 265 270
Pro Met Phe Gln Pro Cys Val Val Pro Pro Lys Pro Trp Thr Gly Ile
275 280 285
Thr Gly Gly Gly Tyr Trp Ala Asn Gly Arg Arg Pro Leu Ala Leu Val
290 295 300
Arg Thr His Ser Lys Lys Ala Leu Met Arg Tyr Glu Asp Val Tyr Met
305 310 315 320
Pro Glu Val Tyr Lys Ala Ile Asn Ile Ala Gln Asn Thr Ala Trp Lys
325 330 335
Ile Asn Lys Lys Val Leu Ala Val Ala Asn Val Ile Thr Lys Trp Lys
340 345 350
His Cys Pro Val Glu Asp Ile Pro Ala Ile Glu Arg Glu Glu Leu Pro
355 360 365
Met Lys Pro Glu Asp Ile Asp Met Asn Pro Glu Ala Leu Thr Ala Trp
370 375 380
Lys Arg Ala Ala Ala Ala Val Tyr Arg Lys Asp Lys Ala Arg Lys Ser
385 390 395 400
Arg Arg Ile Ser Leu Glu Phe Met Leu Glu Gln Ala Asn Lys Phe Ala
405 410 415
Asn His Lys Ala Ile Trp Phe Pro Tyr Asn Met Asp Trp Arg Gly Arg
420 425 430
Val Tyr Ala Val Ser Met Phe Asn Pro Gln Gly Asn Asp Met Thr Lys
435 440 445
Gly Leu Leu Thr Leu Ala Lys Gly Lys Pro Ile Gly Lys Glu Gly Tyr
450 455 460
Tyr Trp Leu Lys Ile His Gly Ala Asn Cys Ala Gly Val Asp Lys Val
465 470 475 480
Pro Phe Pro Glu Arg Ile Lys Phe Ile Glu Glu Asn His Glu Asn Ile
485 490 495
Met Ala Cys Ala Lys Ser Pro Leu Glu Asn Thr Trp Trp Ala Glu Gln
500 505 510
Tyr Ser Pro Phe Cys Phe Leu Ala Phe Cys Phe Glu Tyr Ala Gly Val
515 520 525
Gln His His Gly Leu Ser Tyr Asn Cys Ser Leu Pro Leu Ala Phe Asp
530 535 540
Gly Ser Cys Ser Gly Ile Gln His Phe Ser Ala Met Leu Arg Asp Glu
545 550 555 560
Val Gly Gly Arg Ala Val Asn Leu Leu Pro Ser Glu Thr Val Gln Asp
565 570 575
Ile Tyr Gly Ile Val Ala Lys Lys Val Asn Glu Ile Leu Gln Ala Asp
580 585 590
Ala Ile Asn Gly Thr Asp Asn Glu Val Val Thr Val Thr Asp Glu Asn
595 600 605
Thr Gly Glu Ile Ser Glu Lys Val Lys Leu Gly Thr Lys Ala Leu Ala
610 615 620
Gly Gln Trp Leu Ala Tyr Gly Val Thr Arg Trp Val Thr Lys Arg Ser
625 630 635 640
Val Met Thr Leu Ala Tyr Gly Ser Lys Glu Phe Gly Phe Arg Gln Gln
645 650 655
Val Leu Glu Phe Thr Ile Gln Pro Ala Ile Asp Ser Gly Lys Gly Leu
660 665 670
Met Phe Thr Gln Pro Asn Gln Ala Ala Gly Tyr Met Ala Lys Leu Ile
675 680 685
Trp Glu Ser Val Ser Val Thr Val Val Ala Ala Val Glu Ala Met Asn
690 695 700
Trp Leu Lys Ser Ala Ala Lys Leu Leu Ala Ala Glu Val Lys Asp Lys
705 710 715 720
Lys Thr Gly Glu Ile Leu Arg Lys Arg Cys Ala Val His Trp Val Thr
725 730 735
Pro Asp Gly Phe Pro Val Trp Gln Glu Tyr Lys Lys Pro Ile Gln Thr
740 745 750
Arg Leu Asn Leu Met Phe Leu Gly Gln Phe Arg Leu Gln Pro Thr Ile
755 760 765
Asn Thr Asn Lys Asp Ser Glu Ile Asp Ala His Lys Gln Glu Ser Gly
770 775 780
Ile Ala Pro Asn Phe Val His Ser Gln Asp Gly Ser His Leu Arg Lys
785 790 795 800
Thr Val Val Trp Ala His Glu Lys Tyr Gly Ile Glu Ser Phe Ala Leu
805 810 815
Ile His Asp Ser Phe Gly Thr Ile Pro Ala Asp Ala Ala Asn Leu Phe
820 825 830
Lys Ala Val Arg Glu Thr Met Val Asp Thr Tyr Glu Ser Cys Asp Val
835 840 845
Leu Ala Asp Phe Tyr Asp Gln Phe Ala Asp Gln Leu His Glu Ser Gln
850 855 860
Leu Asp Lys Met Pro Ala Leu Pro Ala Lys Gly Asn Leu Asn Leu Arg
865 870 875 880
Asp Ile Leu Glu Ser Asp Phe Ala Phe Ala
885 890
<210> 40
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Субстрат и т.д.
<400> 40
tttttttttt tttttttttt ttttt 25
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| МУТАЦИИ В ГЕНАХ OAS1 | 2006 |
|
RU2465328C2 |
| НЕ ВСТРЕЧАЮЩИЙСЯ В ПРИРОДЕ ВИРУС РЕПРОДУКТИВНО-РЕСПИРАТОРНОГО СИНДРОМА СВИНЕЙ (BPPCC) И СПОСОБЫ ПРИМЕНЕНИЯ | 2015 |
|
RU2687150C2 |
| ЭКСПРЕССИЯ ПОЛИПЕПТИДОВ НИТРОГЕНАЗЫ В РАСТИТЕЛЬНЫХ КЛЕТКАХ | 2018 |
|
RU2809244C2 |
| Полинуклеотиды, кодирующие релаксин | 2017 |
|
RU2795683C2 |
| ДИСПЛЕЙ ИНТЕГРАЛЬНОГО МЕМБРАННОГО БЕЛКА НА ВНЕКЛЕТОЧНЫХ ОБОЛОЧЕЧНЫХ ВИРИОНАХ ПОКСВИРУСА | 2017 |
|
RU2759846C2 |
| МАТЕРИАЛЫ И МЕТОДЫ, ИСПОЛЬЗУЕМЫЕ ДЛЯ ЛЕЧЕНИЯ РЕСПИРАТОРНЫХ ЗАБОЛЕВАНИЙ У СОБАК | 2020 |
|
RU2811752C2 |
| ВЫДЕЛЕНИЕ НОВОГО ПЕСТИВИРУСА, ВЫЗЫВАЮЩЕГО ВРОЖДЕННЫЙ ТРЕМОР А | 2017 |
|
RU2765658C2 |
| СНИЖЕНИЕ ПРЕВРАЩЕНИЯ НИКОТИНА В НОРНИКОТИН В РАСТЕНИЯХ | 2015 |
|
RU2733837C2 |
| Гибридные белки NPP1 | 2011 |
|
RU2760943C2 |
| СПОСОБЫ ПОЛУЧЕНИЯ ПРОМЫШЛЕННЫХ ПРОДУКТОВ ИЗ РАСТИТЕЛЬНЫХ ЛИПИДОВ | 2015 |
|
RU2743384C2 |
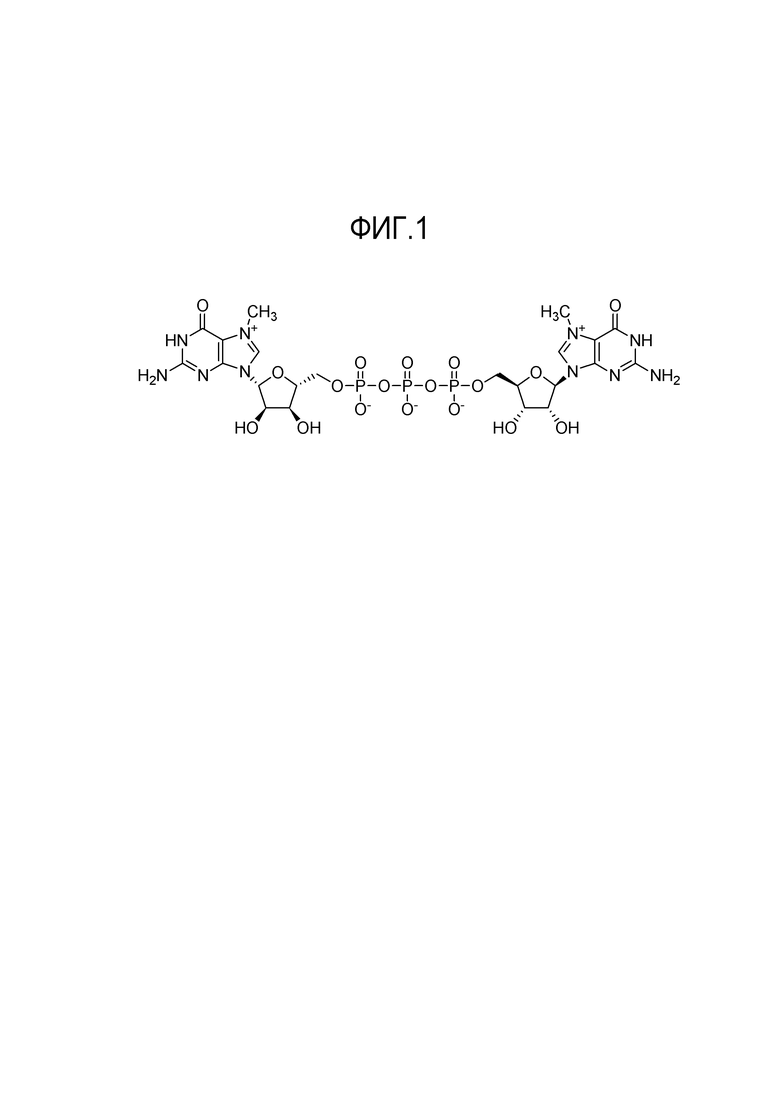
Изобретение относится к биотехнологии и представляет собой сконструированную РНК-полимеразу, содержащую полипептидную последовательность, обладающую по меньшей мере 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичностью последовательности с эталонной последовательностью SEQ ID NO: 4, где указанная полипептидная последовательность содержит по меньшей мере замену 664W/F/Y, 404E/Y, 514F/I/L/Y, 661E/Y, 446W/Y, 136E/I, 137W, 357G/K/L/M/N/Q/R/S/T/V/W, 393L/Y, 394R, 397F/M/Q/W/Y, 401V, 444F/H/l/V, 478F/M/W, 513C/F/K/L/R/T/W/Y, 582N, 635F/R/W, 636L, 637G/P/S, 639H, 642L, 643A, 645V, 653C, 656F/W или 660A/C/M/S/T/W/F/N или любой их комбинации, и где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4 или 15. Изобретение относится также к экспрессирующему вектору и клетке-хозяину для продуцирования сконструированной РНК-полимеразы. Изобретение позволяет получить варианты РНК-полимеразы для селективного включения симметричного кэпированного аналога GTP относительно GTP при инициации транскрипции in vitro. 7 н. и 45 з.п. ф-лы, 1 ил., 7 табл., 7 пр.
1. Сконструированная РНК-полимераза, содержащая полипептидную последовательность, обладающую по меньшей мере 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более идентичностью последовательности с эталонной последовательностью SEQ ID NO: 4, где указанная полипептидная последовательность содержит по меньшей мере замену 664W/F/Y, 404E/Y, 514F/I/L/Y, 661E/Y, 446W/Y, 136E/I, 137W, 357G/K/L/M/N/Q/R/S/T/V/W, 393L/Y, 394R, 397F/M/Q/W/Y, 401V, 444F/H/l/V, 478F/M/W, 513C/F/K/L/R/T/W/Y, 582N, 635F/R/W, 636L, 637G/P/S, 639H, 642L, 643A, 645V, 653C, 656F/W или 660A/C/M/S/T/W/F/N или любой их комбинации, и где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4 или 15.
2. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 664W/F/Y, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
3. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 404E/Y, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
4. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 514F/I/L/Y, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
5. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 661E/Y, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
6. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 446W/Y, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
7. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 136E/I, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
8. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 137W, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
9. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 357G/K/L/M/N/Q/R/S/T/V/W, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
10. Сконструированная РНК-полимераза по п.9, где указанная полипептидная последовательность содержит по меньшей мере замену 357R, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
11. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 393L/Y, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
12. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 394R, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
13. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 397F/M/Q/W/Y, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
14. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 401V, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
15. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 444F/H/l/V, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
16. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 478F/M/W, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
17. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 513C/F/K/L/R/T/W/Y, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
18. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 582N, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
19. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 635F/R/W, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
20. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 636L, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
21. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 637G/P/S, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
22. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 639H, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
23. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 642L, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
24. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 643A, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
25. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 645V, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
26. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 653C, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
27. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 656F/W, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
28. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 660A/C/M/S/T/W/F/N, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
29. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену 664W, 357R, 394R, 404Y, 514L/I/F, 661E, 446W или 136I или любую их комбинацию, где положения аминокислот указанной полипептидной последовательности пронумерованы относительно SEQ ID NO: 4.
30. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену или группу замен 397F/513W/635W, 397F/513Y/635W, 397W/513Y/635W, 397W/513W/635W, 397W/513Y/635W/660F, 397W/513W/635W/660W, 397Y/513W/635W/660F, 475V/513W/635W/660Y, 513F/660W/664Y, 513W/635W/660F, 513W/635W/664W, 513Y/635W/660F, 513Y/635W/660Y, 513Y/660W/664W или 660Y/664W, где положения аминокислот пронумерованы относительно SEQ ID NO: 4.
31. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену или группу замен 397F, 397F/513F, 397F/513F/635F, 397F/513F/635W, 397F/513F/635W/660W, 397F/513W/635W, 397F/513W/635W/656F/664F, 397F/513W/664W, 397F/513Y, 397F/513Y/635W, 397F/513Y/635W/656W, 397F/513Y/635W/664F, 37F/513Y/656H/660W, 397F/635F/660W, 397F/635W, 397F/660W, 397F/664W, 397W, 397W/513F, 397W/513F/635F/656F/660F/664W, 397W/513F/635F/660W, 397W/513F/635W, 397W/513F/660F, 397W/513F/660W, 397W/513F/660Y/664F, 397W/513W, 397W/513W/635F, 397W/513W/635F/656W/660F, 397W/513W/635W, 397W/513W/635W/656Y, 397W/513W/635W/660W, 397W/513W/635W/660W/664Y, 397W/513W/635W/660Y/664Y, 397W/513W/660W, 397W/513W/660W/664Y, 397W/513W/664F, 397W/513W/664W, 397W/513Y/635F, 397W/513Y/635W/656Y/660W/664W, 397W/513Y/635W, 397W/513Y/635W/656Y/664Y, 397W/513Y/635W/660F, 397W/635F, 397W/635F/664F, 397W/635W, 397W/635W/656F/664F, 397W/635W/656F/664W, 397W/635W/656F/664Y, 397W/635W/660W, 397W/635W/660Y, 397W/635W/664F, 397W/635W/664W/850T, 397W/660F/664F, 397W/660F/664Y, 397W/660W, 397W/837K, 397Y, 397Y/513F/635W/656F/660W/664F, 397Y/513F/635W/664W, 397Y/513W/635F/664Y, 397Y/513W/635W/660F, 397Y/513W/656W/660W, 397Y/513Y, 397Y/513Y/635W, 397Y/635F, 397Y/635F/660W, 397Y/635W, 397Y/635W/656F/660Y/664W, 397Y/635W/660F, 397Y/660F/664W, 397Y/664F, 399E/635F/660W, 475V/513W/635W/660Y, 513F, 513F/635F, 513F/635W, 513F/635W/656W, 513F/635W/664W, 513F/660W, 513F/660W/664F, 513F/660W/664Y, 513W, 513W/635F, 513W/635W, 513Y/635F/660F/664Y, 513W/635W/656W/660F, 513W/635W/660F, 513W/635W/664W, 513W/656W/664W, 513W/656Y/660W, 513W/660F, 513W/660W, 513Y/635F, 513Y/635F/664W, 513Y/635R/656F/664Y, 513Y/635W, 513Y/635W/660F, 513Y/635W/660Y, 513Y/635W/660Y/664F, 513Y/660W, 513Y/660W/664W, 513Y/660Y/664F, 513Y/664Y, 635F/656F/664Y, 635F/656Y/664W, 635F/660W/664F, 635W, 635W/656W, 635W/660F, 635W/660W, 635W/664W, 656W/660F/664Y, 656W/660W/664Y, 658P, 660F, 660F/664F, 660F/664Y, 660W/664F, 660Y/664W, 664F или 664W, где положения аминокислот пронумерованы относительно SEQ ID NO: 4.
32. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит по меньшей мере замену или группу замен 113M/137W/513R, 136E/357I/404Y/514I, 136I/357I/514F, 136I/357K/514F, 136I/401V, 136I/401V/404Y, 137W/401I, 137W/401S, 137W/401S/513R, 137W/401S/513W, 137W/401V, 137W/513W, 137W/513R/621R, 357N/394R/446W/514I, 357R/394R/401V/404Y/514L, 357R/514F, 401S/513R/635W, 401S/635W, 401V/404Y, 401V/404Y/514L, 513L/635W, 513L/660W, 513R/635W/656F, 635W/660T, 660S/T, где положения аминокислот пронумерованы относительно SEQ ID NO: 15.
33. Сконструированная РНК-полимераза по п.1, где указанная сконструированная РНК-полимераза представляет собой вариант сконструированной полимеразы, приведенный в таблице 5.3, 5.4, 5.5 и/или 5.6.
34. Сконструированная РНК-полимераза по п.1, где указанная полипептидная последовательность содержит SEQ ID NO: 15, 17, 19, 21, 23, 25, 27, 29, 31, 33, 35, 37 или 39.
35. Сконструированная РНК-полимераза по любому из пп.1-34, где указанная сконструированная полимераза имеет по меньшей мере одно улучшенное свойство по сравнению с РНК-полимеразой T7 дикого типа.
36. Сконструированная РНК-полимераза по п.35, где указанное по меньшей мере одно улучшенное свойство выбрано из увеличенной селективности в отношении аналога кэпа относительно GTP в ходе инициации трансляции, увеличенной экспрессии белка, увеличенной стабильности в буфере для хранения и увеличенной стабильности в условиях реакции.
37. Сконструированная РНК-полимераза по любому из пп.1-36, где полимераза сохраняет выход РНК, точность транскрипции, термостабильность, экспрессию белка, стабильность при -20°C или стабильность в условиях реакции, эквивалентные РНК-полимеразе T7 дикого типа.
38. Сконструированная РНК-полимераза по любому из пп.1-37, где указанная сконструированная полимераза является очищенной.
39. Полинуклеотид, кодирующий по меньшей мере одну сконструированную РНК-полимеразу по любому из пп.1-37.
40. Полинуклеотид по п.39, где указанный полинуклеотид функционально связан с контрольной последовательностью.
41. Полинуклеотид по п.39 или 40, содержащий полинуклеотидную последовательность, которая является кодон-оптимизированной.
42. Экспрессирующий вектор, содержащий по меньшей мере один полинуклеотид по любому из пп.39-41.
43. Клетка-хозяин для продуцирования сконструированной РНК-полимеразы по любому из пп.1-37, содержащая по меньшей мере один экспрессирующий вектор по п.42.
44. Клетка-хозяин по п.43, где указанная клетка является прокариотической клеткой.
45. Способ продуцирования сконструированной РНК-полимеразы в клетке-хозяине, включающий культивирование клетки-хозяина по п.43 или 44 в подходящих условиях культивирования, так чтобы продуцировалась по меньшей мере одна сконструированная РНК-полимераза.
46. Способ по п.45, дополнительно включающий выделение по меньшей мере одной сконструированной РНК-полимеразы из культуры и/или клетки-хозяина.
47. Способ по п.45 или 46, дополнительно включающий стадию очистки указанной по меньшей мере одной сконструированной РНК-полимеразы.
48. Композиция для продуцирования РНК-транскрипта, содержащая по меньшей мере одну сконструированную РНК-полимеразу по любому из пп.1-38 и по меньшей мере ДНК-матрицу.
49. Способ продуцирования кэпированных РНК-транскриптов, включающий предоставление композиции, содержащей: i) по меньшей мере одну сконструированную РНК-полимеразу по любому из пп.1-38, динуклеотидный аналог кэпа, и ii) ДНК-матрицу; воздействие на указанную ДНК-матрицу указанной композицией в условиях, чтобы указанная сконструированная РНК-полимераза продуцировала кэпированный РНК-транскрипт.
50. Способ по п.49, где динуклеотидный аналог кэпа представляет собой альфа, гамма-бис(N7-метилгуанозин)трифосфат (m7G(5')ppp(5')m7G) или антиреверсный аналог кэпа 3'-O-Me-m7G(5')ppp(5')G.
51. Способ по п.49 или 50, где динуклеотидный аналог кэпа представляет собой альфа, гамма-бис(N7-метилгуанозин)трифосфат.
52. Способ по любому из пп.49-51, дополнительно включающий добавление неорганической пирофосфатазы.
| ПОДБОРЩИК ПЛОДОВ БАХЧЕВЫХ КУЛЬТУР | 2006 |
|
RU2316934C1 |
| СПОСОБ ПРОИЗВОДСТВА КОМПОТА ИЗ ВИНОГРАДА | 2009 |
|
RU2377938C1 |
| US 20130224793 A1, 29.08.2013 | |||
| ПРОСТОЙ, ЭФФЕКТИВНЫЙ И БЫСТРЫЙ СПОСОБ ФЕРМЕНТАТИВНОЙ МОДИФИКАЦИИ И СИНТЕЗА НУКЛЕИНОВОЙ КИСЛОТЫ IN VITRO С ИСПОЛЬЗОВАНИЕМ МИКРОВОЛНОВОГО ИЗЛУЧЕНИЯ | 2002 |
|
RU2295568C2 |
Авторы
Даты
2023-05-16—Публикация
2018-06-19—Подача