ОБЛАСТЬ ТЕХНИКИ
Изобретение относится к области поиска терапевтически значимых молекулярных мишеней для заболеваний путем применения методов машинного обучения к комбинированным данным, включающим графы сигнальных путей, омиксные и текстовые типы данных, в частности к компьютерно-реализуемому способу предсказания и приоритизации лекарственных мишеней для заболеваний на основе болезнь-специфичного графа с помощью методов машинного обучения.
Представленное решение может быть использовано, по меньшей мере, для разработки новых эффективных лекарств.
УРОВЕНЬ ТЕХНИКИ
Разработка лекарства является трудоемким, дорогостоящим и сложным процессом, в котором есть два основных этапа, предшествующие доклиническим и клиническим исследованиям. Первый — это идентификация, приоритизация и валидация мишени с подходящими физическими свойствами, модуляция которых может повлиять на метаболические пути, ассоциированные с болезнью. Вторым этапом является выявление и оптимизация соединений, которые связываются с мишенью и модулируют ее биологическую активность. Оба этапа являются принципиально важными для разработки новых эффективных лекарств. Тем не менее, многие лекарства-кандидаты до сих пор терпят неудачу в клинических исследованиях из-за низкой эффективности, связанной с неправильным выбором мишени. В то время как вычислительные методы для прогнозирования связывания молекул-кандидатов с белком-мишенью достаточно хорошо развиты, методы компьютерной приоритизации мишеней остаются в меньшей степени разработанными, поэтому традиционно ученые определяли мишени путем поиска через соответствующую литературу, а также на основе анализа экспрессии белка и мРНК, интегрируя данные экспрессии с анализом путей, экспериментируя с нокаут-мышами, исследуя соматические мутации, вариации числа копий генов, а также используя накопленные знания из множественных экспериментальных исследований для создания гипотезы о том, как белки или другие макромолекулы могут работать в качестве мишеней. Однако, ручная интерпретация многих источников данных склонна к предвзятому определению мишеней, поскольку это ограничивает возможности использования всех доступных данных. Путем вычислительной интеграции многочисленных источников биологических данных для анализа предшествующих знаний можно заметно ускорить процесс идентификации терапевтических мишеней. Таким образом, вычислительные подходы к поиску мишеней в большинстве случаев должны быть направлены на ранжирование белков на основании их вероятности быть мишенью в контексте конкретного заболевания.
Ниже перечислены основные известные на сегодняшний день методы поиска молекулярных мишеней для заболеваний.
В статье [1] описан подход Emig et al. Авторы представляют сетевой подход для прогнозирования мишеней лекарств для заболеваний. Метод позволяет как репозиционировать известные для других заболеваний лекарственные мишени на конкретное заболевание, так и прогнозировать неиспользованные лекарственные мишени, которые не используются для лечения какого-либо заболевания. В данном подходе авторы используют графы белок-белковых взаимодействий, вычисляют свойства вершин графов, получают сигнатуры дифференциально экспрессируемых генов, объединяют сигнатуры дифференциальной экспрессии и свойств вершин графов, обучают модели логистической регрессии на полученной матрице и выводят приоритетный список мишеней для лекарств.
В статье [2] описан подход Guiltytargets. В статье использовались графы белок-белковых взаимодействий (STRING и HIPPIE) в сочетании с информацией об дифференциально экспрессируемых генах для предсказания молекулярных мишеней для разработки лекарств. Авторы решают задачу классификации узлов с помощью графа на основе белок-белковых взаимодействий, разметки графа данными по дифференциальной экспрессии генов, свёртки графа на основе простой модели, тренировки классификационной модели в новом пространстве.
Однако в данных подходах отсутствует одновременное использование данных о дифференциальной экспрессии генов и текстовой информации о мишенях и заболеваниях для разметки графов на основе белок-белковых взаимодействий, а также валидация модели машинного обучения для предсказания и приоритизации лекарственных мишеней по времени (валидация по времени «машина времени»).
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Техническим результатом заявляемого изобретения является автоматическое ранжирование списка белок-кодирующих генов с точки зрения вероятности быть лекарственной мишенью для заболеваний с помощью методов машинного обучения на основе болезнь-специфичного графа, что повышает точность прогнозирования вероятности белок-кодирующих генов быть лекарственными мишенями для заболеваний, ускоряет процесс идентификации лекарственных мишеней для заболеваний, и приводит к ускорению разработки новых эффективных лекарств.
Указанный технический результат достигается за счёт того, что:
В компьютерно-реализуемом способе предсказания и приоритизации лекарственных мишеней для заболеваний на основе болезнь-специфичного графа с помощью методов машинного обучения на вычислительном устройстве, содержащем процессор и память, хранящую исполняемые процессором инструкции, исполняют инструкции, включающие этапы, на которых:
создают граф белок-белковых взаимодействий, в котором узлами являются белок-кодирующие гены, ребрами являются взаимодействия белок-кодирующих генов;
создают болезнь-специфичный граф с помощью разметки графа белок-белковых взаимодействий с помощью болезнь - специфической информации, включающей текстовые и омиксные данные;
с помощью сверточных нейронных сетей осуществляют свертку полученного болезнь-специфичного графа в векторное представление, и для каждого узла/белок-кодирующего гена получают вектор, который характеризует топологические свойства болезнь-специфичного графа и данные дифференциальной экспрессии для заболеваний;
подают полученное векторное представление болезнь-специфичного графа в обученную модель машинного обучения для предсказания и приоритизации лекарственных мишеней и на выходе указанной модели получают ранжированный список белок-кодирующих генов с точки зрения вероятности быть лекарственной мишенью для заболеваний.
В способе может быть осуществлена разметка графа белок-белковых взаимодействий на основе данных о дифференциальной экспрессии генов, данных о связях заболеваний с лекарственными мишенями, данных об онтологии заболеваний, данных о лекарственных мишенях, данных о свойствах белков путем добавления в граф белок-белковых взаимодействий следующих связей: белок-кодирующий ген - болезнь, белок-кодирующий ген - лекарственная мишень, болезнь-лекарственная мишень, болезнь-болезнь.
В способе модель машинного обучения для предсказания и приоритизации лекарственных мишеней может быть валидирована при помощи методов обогащения топ К и статистически - гипергеометрического теста.
В способе модель машинного обучения для предсказания и приоритизации лекарственных мишеней может быть валидирована по времени.
В способе для валидации модели машинного обучения для предсказания лекарственных мишеней по времени может быть использован обучающий набор, полученный на основе данных, которые были известны до заданной временной точки, и тестовый набор, полученный на основе данных, которые стали известны после указанной заданной временной точки.
В способе может быть использована строгая и нестрогая стратегия для получения обучающего набора, полученного на основе данных, которые были известны до заданной временной точки, и тестового набора, полученного на основе данных, которые стали известны после указанной заданной временной точки.
ОПИСАНИЕ ЧЕРТЕЖЕЙ
Реализация изобретения будет описана в дальнейшем в соответствии с прилагаемыми чертежами, которые представлены для пояснения сути изобретения и никоим образом не ограничивают область изобретения.
Заявляемое изобретение проиллюстрировано фигурами 1-4, на которых изображены:
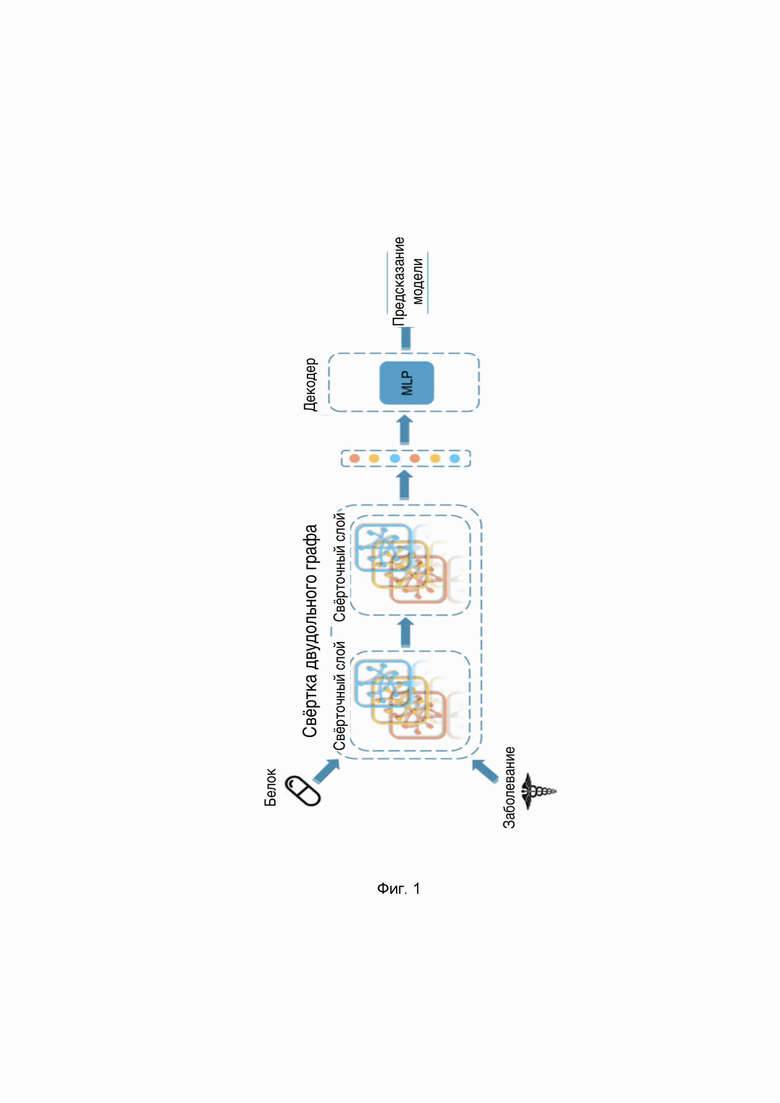
Фиг. 1 – иллюстрирует пример общей схемы компьютерно-реализуемого способа предсказания и приоритизации лекарственных мишеней для заболеваний на основе болезнь-специфичного графа с помощью методов машинного обучения.
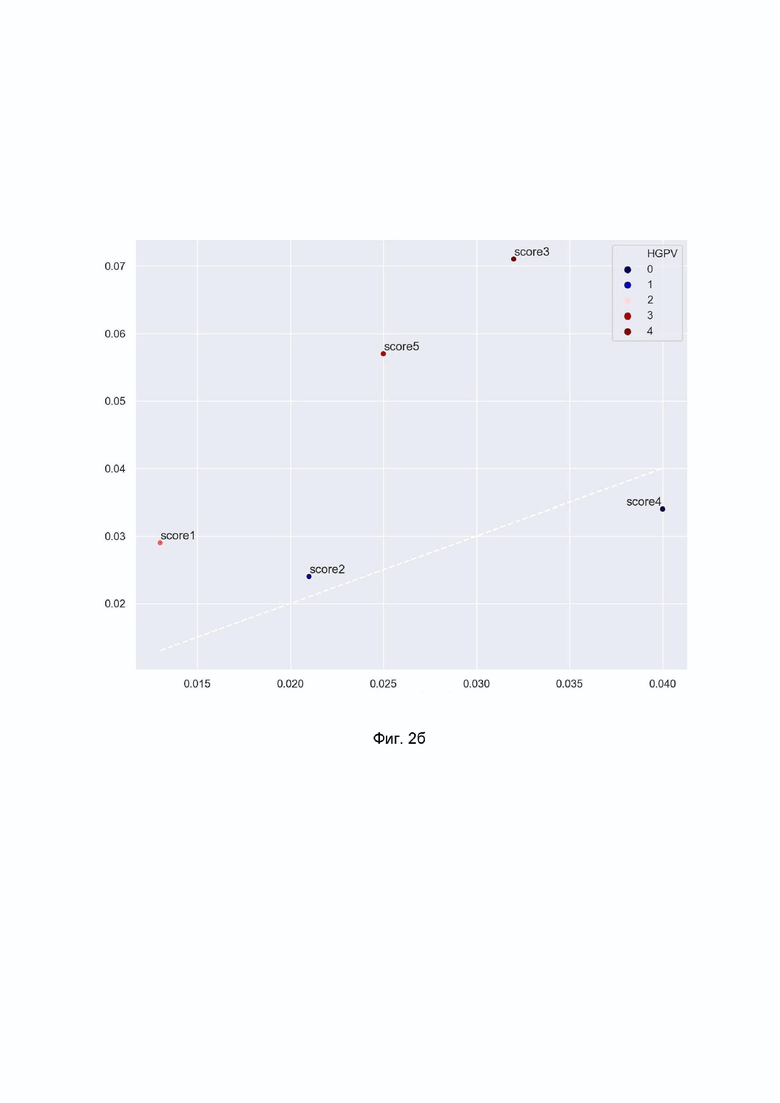
Фиг. 2а, 2б – иллюстрируют примеры графиков визуализации результатов валидации модели машинного обучения предсказания и приоритизации лекарственных мишеней.
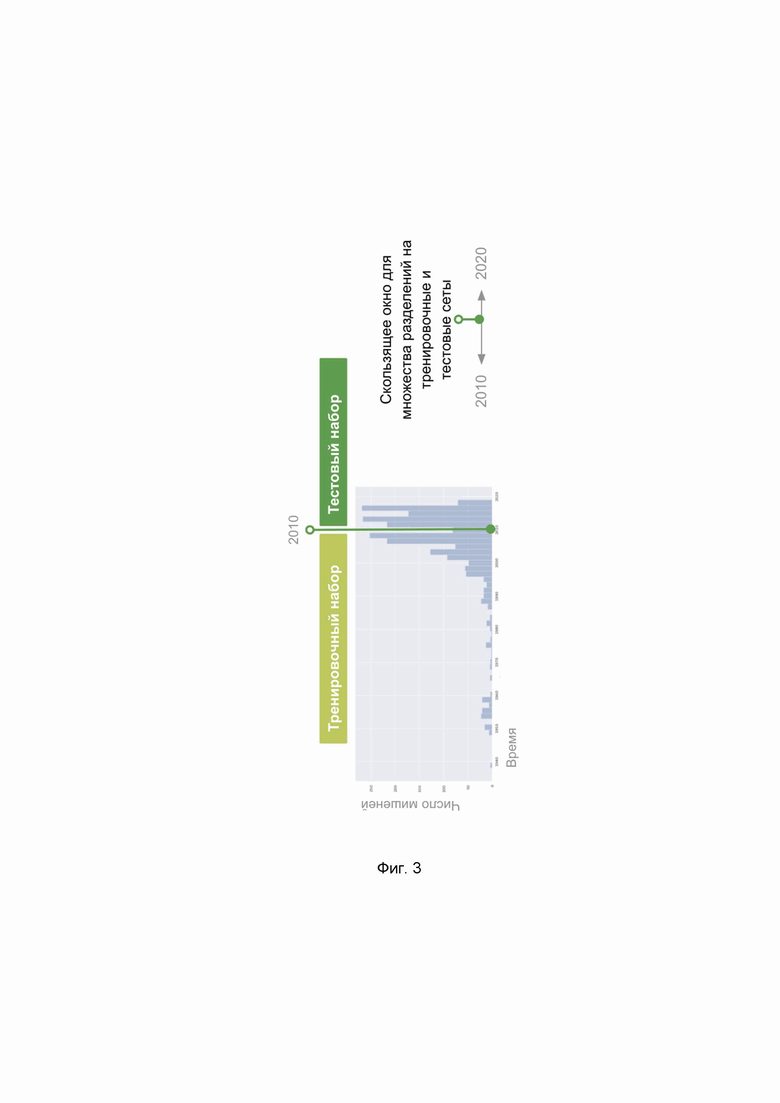
Фиг. 3 – иллюстрирует пример валидации по времени модели машинного обучения предсказания и приоритизации лекарственных мишеней.
Фиг. 4 – иллюстрирует общую схему вычислительного устройства для реализации компьютерно-реализуемого способа предсказания и приоритизации лекарственных мишеней для заболеваний на основе болезнь-специфичного графа с помощью методов машинного обучения.
ДЕТАЛЬНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
В приведенном ниже подробном описании реализации изобретения приведены многочисленные детали реализации, призванные обеспечить отчетливое понимание настоящего изобретения. Однако, квалифицированному в предметной области специалисту будет очевидно, каким образом можно использовать настоящее изобретение, как с данными деталями реализации, так и без них. В других случаях хорошо известные методы, процедуры и компоненты не были описаны подробно, чтобы не затруднять излишне понимание особенностей настоящего изобретения.
Кроме того, из приведенного изложения будет ясно, что изобретение не ограничивается приведенной реализацией. Многочисленные возможные модификации, изменения, вариации и замены, сохраняющие суть и форму настоящего изобретения, будут очевидными для квалифицированных в предметной области специалистов.
Наиболее перспективные современные подходы к поиску мишеней для заболеваний используют несколько источников информации, как например граф белок-белковых взаимодействий, дифференциальное изменение экспрессии гена, биологические роли самих генов, текстовую информацию и т.д. При этом использование комплексной информации для обучения алгоритмов классификации терапевтических мишеней технически сложнее, поскольку информация из разных источников имеет разнообразный формат и размерность. Для решения этой проблемы часто используют эмбеддинги (векторное представление) или алгоритмы свертки, позволяющие преобразовать информацию в более удобный формат. Например, есть алгоритмы графовых сверток, в большинстве своем основанные на свойствах узлов, случайных блужданиях по графу или на применении графовых нейронных сетей. После структуризации информации становится возможным применять разные методы машинного обучения для классификации узлов на мишени и не мишени.
В настоящем решении для построения болезнь-специфичного графа используется граф белок - белковых взаимодействий. Граф белок-белковых взаимодействий (граф сигнальных путей) необходим для алгоритмов распространения сигнала [3]. Предпочтительно в качестве графа белок - белковых взаимодействий использовать STRING, который агрегирует информацию о прямых физических и косвенных взаимодействиях белков рассматриваемого живого организма, например человека, животного. Все взаимодействия объединены в сеть/ненаправленный граф, где узлами являются белок-кодирующие гены, ребрами являются взаимодействия белок-кодирующих генов. Каждая связь имеет рассчитанный вес, который отображает уровень достоверности установления этой связи. Данные для расчёта веса (скора) загружают из базы STRING, вес рассчитывается на основе количества источников, подтверждающих данное белок-белковое взаимодействие. Например, если есть подтверждения высокопроизводительных методов, описание в литературе и подтверждение точечными экспериментами, то вес будет максимальным. Например, вес отображает уровень достоверности установления связи белок-кодирующих генов от 0 до 1000. Данный вес рассчитывается на основании 7 подвесов, 3 из которых рассчитывается на основании геномных данных, и по одному на основе данных: коэкспрессии, текст-майнинга на основе научных публикаций, биохимических экспериментов, курируемых баз данных биологических путей и макромолекулярных комплексов.
Для разметки узлов и построения связей болезнь-специфичного графа на основе графа белок - белковых взаимодействий используют следующие биологические данные:
- белок-белковые взаимодействия;
- данные о связях белок-кодирующих генов с заболеваниями;
- данные о лекарственных (терапевтически значимых) мишенях;
- данные дифференциальной экспрессии генов;
- данные об онтологии заболеваний;
- данные о связях болезней с лекарственными (терапевтически значимыми) мишенями;
- свойства белков.
Биологические данные для построения болезнь-специфичного графа извлекают из различных источников данных. В Таблице 1 приведены примеры таких источников данных, а также примеры извлекаемых данных и их использования.
Таблица 1
Текстовые данные
В качестве источника данных о связях болезней с лекарственными мишенями на основании текстовых данных использовалась база https://pharmacognitive.com/. База собирается в результате поиска именованных сущностей (названий генов, заболеваний) в абстрактах биомедицинских статей. На основании полученных статистик вычисляются веса для связей белок-кодирующий ген-болезнь, используемые в финальной модели.
Данные дифференциальной экспрессии генов
Данные о дифференциальной экспрессии генов получают из GEO. Для каждой болезни получают список достоверно отличающихся генов при сравнении нормальных и патологических образцов на основании следующих критериев: pValue(FDR) < 0.1, abs(log2FoldChange) > 1. Добавляют в болезнь-специфичный граф связи белок-кодирующий ген – болезнь из списка отличившихся.
Таблица 2 показывает, для какого количества заболеваний может быть построена модель и на каком количестве датасетов на основании данных из Пандомикс.
Таблица 2
Информация о лекарственных мишенях
В изобретении используются данные о лекарственных мишенях из двух открытых баз данных: Chembl и ClinTrials. В Таблице 3 приведены данные о количестве уникальных болезней и уникальных мишеней, полученных и указанных ба данных.
Таблица 3
Таким образом, для предсказания и приоритизации лекарственных мишеней для заболеваний создают граф белок-белковых взаимодействий, в котором узлами являются белок-кодирующие гены, ребрами являются взаимодействия белок-кодирующих генов. Создают болезнь-специфичный граф с помощью комбинированных данных, включающих граф сигнальных путей, омиксные и текстовые типы данных, путем разметки графа белок-белковых взаимодействий (графа сигнальных путей) с помощью болезнь - специфической информации, включающей текстовые и омиксные данные.
Осуществляют разметку графа белок-белковых взаимодействий на основе данных о дифференциальной экспрессии генов, данных о связях заболеваний с лекарственными мишенями, данных об онтологии заболеваний, данных о лекарственных мишенях, данных о свойствах белков путем добавления в граф белок-белковых взаимодействий следующих связей: белок-кодирующий ген - болезнь, белок-кодирующий ген - лекарственная мишень, болезнь-лекарственная мишень, болезнь-болезнь. Вес для связи связи белок-кодирующий ген - болезнь рассчитывают на основе количества факторов, подтверждающих связь (факторы - GWAS, дифференциальная экспрессия, наличие известного лекарственного средства). Связь белок-кодирующий ген - лекарственная мишень является не взвешенной. Связь болезнь-лекарственная мишень - не взвешенная, 0 либо 1 в зависимости от применения лекарственной мишени для терапии заболевания. Вес связи болезнь-болезнь определяется на основе расстояния на графе онтологии заболеваний.
Например, основываясь на омиксных данных дифференциальной экспрессии, полученных из открытой базы данных OpenTargets и текстовой информации, полученной из pubmed, размечают узлы графа белок-белковых взаимодействий из базы данных STRING, получив тем самым болезнь-специфичный граф.
С помощью сверточных нейронных сетей осуществляют свертку полученного болезнь-специфичного графа в векторное представление, и для каждого узла/белок-кодирующего гена получают вектор, который характеризует топологические свойства болезнь-специфичного графа и данные дифференциальной экспрессии для заболеваний.
Например, топологическую информацию из размеченного графа переводят в векторное пространство, используя алгоритм “MAGNN”, основанный на сверточных нейронных сетях [4]. Данный алгоритм показал наилучшие результаты метрик в сравнительном анализе с другими алгоритмами [5].
В настоящем изобретении модель тестируется на стандартной размерности вектора (эмбеддинга) 64, что является распространенной размерностью по умолчанию. Тем не менее, размерность вектора можно менять с целью найти оптимальный баланс между качеством и скоростью. Очень маленький вектор может не вмещать всю полезную информацию, которую можно извлечь из графа. Увеличение вектора выше оптимального размера существенно снижает производительность системы, поскольку приводит к более тяжелым расчетам, и при этом не хранит полезной информации сверх определенного лимита. Размерность 64 выбрана на основании того, что является сбалансированной.
Далее подают полученное векторное представление болезнь-специфичного графа в обученную модель машинного обучения для предсказания и приоритизации лекарственных мишеней и на выходе указанной модели получают ранжированный список белок-кодирующих генов с точки зрения вероятности быть лекарственной мишенью для заболеваний. На Фиг. 1 представлен пример общей схемы компьютерно-реализуемого способа предсказания и приоритизации лекарственных мишеней для заболеваний на основе болезнь-специфичного графа с помощью методов машинного обучения.
В тренировочной выборке все известные лекарственные мишени размечают как положительный класс, а остальные белок-кодирующие гены размечают как вероятно положительный класс. Используют данные об известных лекарственных мишенях из баз данных Therapeutic Target Database (TTD) и Open Targets Platform (OpenTargets). После этого проводится обучение модели машинного обучения для предсказания и приоритизации лекарственных мишеней, например, классификатора на основе линейной регрессии. При этом модель может меняться в зависимости от качества, показанного на валидации. Полученная модель валидируется при помощи методов обогащения топК и статистически - гипергеометрического теста. После подбора гиперпараметров модели и получение хороших результатов валидации модель готова к предсказанию и приоритизации новых лекарственных мишеней заболеваний. Процесс подбора происходит с помощью оптимизации гиперпарамеров поиском по решётке.
Описание методики валидации
Для валидации использовались известные лекарственные мишени из открытых баз данных TTD и Opentargets.
Обогащение топовых К мишеней - статистически - гипергеометрический тест.
Человек не в состоянии рассмотреть все значения метрики для 24 тысяч белок-кодирующих генов, поэтому анализируют способность модели предсказания выводить релевантные гены в определенное количество лучших (верхних) при ранжировании по исследуемой метрике для предсказания мишеней. Поскольку классические метрики оценки качества ранжирования требуют эталонного ранжирования для сравнения, что редко возможно при оценке известных белков-мишеней для лекарства, а метрики оценки качества классификации плохо подходят к настоящей задаче и часто слабо устойчивы к малому количеству образцов из положительного класса (в настоящем решении - известных молекулярных мишеней для болезни), были разработаны собственные подходы к валидации моделей, которые не используют стандартные метрики. Для оценки качества работы модели (по сравнению со случайной) используется гипергеометрическое распределение. Распределение параметризуется значением k, которое определяет количество верхних значений анализируемого скора, в пределах которого ищут молекулярные мишени. В верхних k генах подсчитывается количество уже известных мишеней, после чего вычисляется значение p-value, указывающее вероятность получения данного или большего числа мишеней путем случайного взятия k генов из генофонда без замены. В итоге данная метрика опирается на статистическую значимость обогащения заданного количества наиболее значимых генов мишенями по сравнению со случайным попаданием мишеней в топ.
Обогащение топК - количественно - log отношения
Для оценки силы эффекта используется следующая формула:
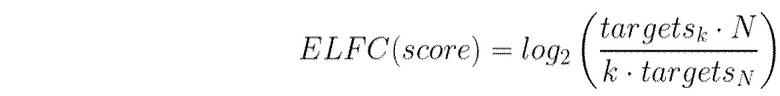 ,
,
где targetsk - количество известных мишеней в верхних k генах при ранжировании по убыванию скора,
N - общее количество генов, для которых был посчитан скор,
k - тестируемое количество верхних генов при ранжировании по убыванию скора,
targetsN - общее количество мишеней во всех генах, для которых рассчитан скор.
Для оценки значимости эффекта используется формула:
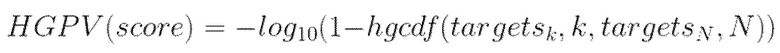 ,
,
где hgcdf означает значение p-value гипергеометрического теста, принцип которого описан выше.
Для анализа предварительно фиксируется значение k, по умолчанию используется обогащение в верхних 100 генах. Для визуализации результатов используются графики, где по оси X отложено значение силы эффекта, а по Y - его статистическая значимость. Пример графика приведен на Фиг. 3. Скоры в верхнем правом углу (score1, score3, score5) лучше всех справляются с поиском известных мишеней, в то время как в левом нижнем углу (score2, score4) находятся те, ранжирование которых слабее всего отличается от случайного угадывания.
На приведенном на Фиг. 2б графике базовый процент мишеней для генов с рассчитанной оценкой отображается по оси X, а результирующий процент мишеней в пределах верхних k отображается по оси Y. Пунктирная линия соответствует ситуации X = Y, то есть доля мишеней в верхних k равняется доле мишеней в фоне. Чем выше точка относительно линии, тем сильнее обогащение в топе по сравнению с фоном. При этом статистическая значимость отклонения показана цветом точек (синий = низкая значимость, красный = высокая значимость).
Валидация по времени
Валидация на уже известных мишенях не дает представления о способности алгоритма находить новые мишени. Для решения этой проблемы используют подход, который характеризуют как "машина времени". Для обучения алгоритма используются только данные, которые были известны до заданной временной точки, а при валидации проверяется обогащение выдачи высоких скоров мишенями, которые были открыты после этой временной точки. Это позволяет оценить перспективность модели для предсказания принципиально новых молекулярных мишеней для заболеваний.
Количество новых уникальных генов-молекулярных мишеней для лекарств в каждый год отображено на Фиг. 3. Выставляя год отсечки можно настраивать количество данных для обучения и тестирования моделей, в результате можно будет оценить способность модели предсказывать гены, которые ранее не были использованы в клинических исследованиях, но имеют хорошую связь с заболеванием и позднее были выбраны в качестве мишени. Устанавливая временную отсечку, можно найти модель, лучше всего умеющую находить гены, которые с хорошим шансом станут лекарствами в перспективе последующих лет (Фиг.3).
При разбиении связей на обучающую (тренировочную) и тестовую с помощью «машины времени» связь может попасть только в одну из выборок: обучающую или тестовую, при этом эти выборки никогда не пересекаются. При этом возможно использование двух стратегий: нестрогой и строгой. При нестрогой стратегии связи ген-болезнь делятся четко по дате возникновения без каких-либо дополнительных проверок. При строгой стратегии если лекарство связывает 50% или более уже известных в контексте данной болезни белковых мишеней и при этом задевает лишь несколько новых, то считают, что оно не приносит ничего нового, в результате чего все его таргеты помещаются в тестовую выборку. Такое часто бывает в случае, если лекарство действует на белковый комплекс и ранее было воздействие на один его участок, например, на белки prot1, prot2, prot4, prot6, а позднее появилось лекарство, в аннотацию которого добавили все белки комплекса - в нашем примере это также prot3 и prot5. Этот подход позволяет избежать "утечки" известных данных в тестовую выборку, но при этом может также отфильтровать лекарства, которые помимо известных механизмов затрагивают какие-то новые. К тому же строгая стратегия существенно уменьшает объем тестовой выборки и для поздних отсечек при данном подходе для многих болезней крайне мало данных для тестирования. При любой из вышеперечисленных стратегий из тестировочной части выборки при необходимости часть данных отводится в отдельную валидационную группу.
Предназначение модели - ранжирование списка генов с точки зрения вероятности быть мишенью для различных заболеваний.
Ниже приведена Таблица 4 с использованными транскриптомными данными. Это данные об экспрессии по двум болезням, выбранным в качестве примера - диабет второго типа [6] и ожирение [7].
Таблица 4
В Таблице 5 приведены топ 20 таргетов (лекарственных мишеней), полученных при помощи вышеописанной модели для двух выбранных заболеваний: диабета второго типа и ожирения при обучении на данных до 2019 года и тестировании на данных после 2019 года.
Таблица 5
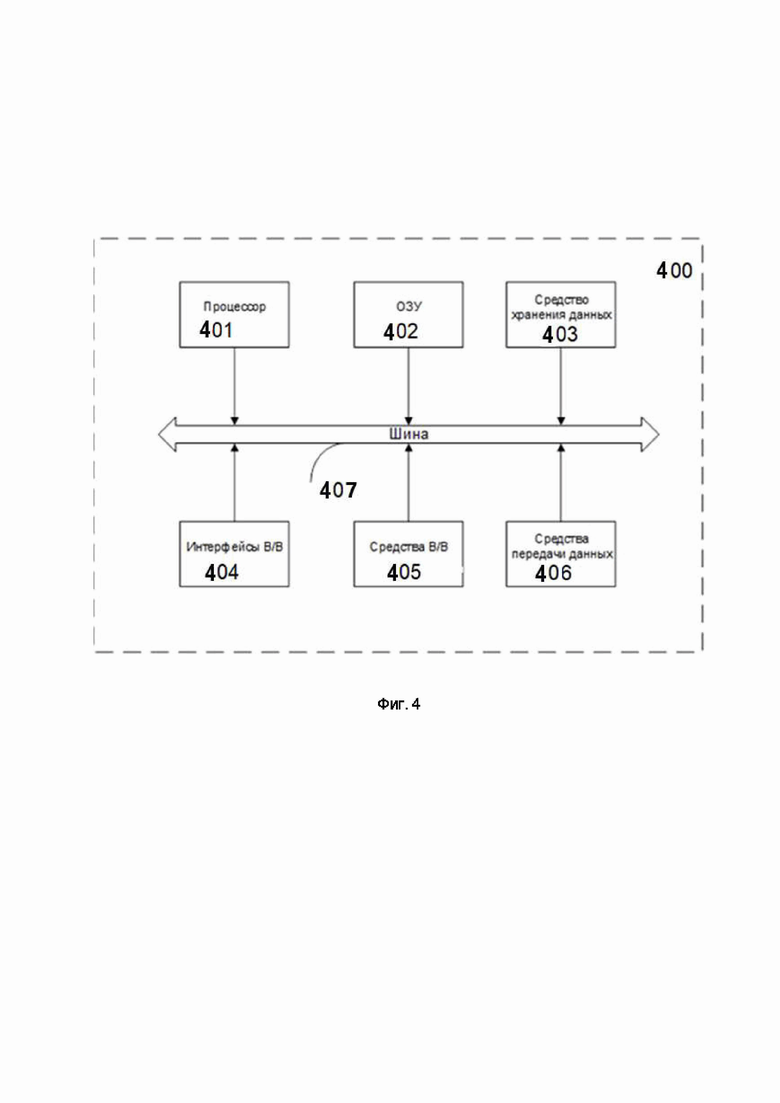
На Фиг. 4 представлена общая схема вычислительного устройства (400), обеспечивающего обработку данных, необходимую для реализации заявленного решения.
В общем случае устройство (400) содержит такие компоненты, как: один или более процессоров (401), по меньшей мере одну память (402), средство хранения данных (403), интерфейсы ввода/вывода (404), средство В/В (405), средства сетевого взаимодействия (406).
Процессор (401) устройства выполняет основные вычислительные операции, необходимые для функционирования устройства (400) или функциональности одного или более его компонентов. Процессор (401) исполняет необходимые машиночитаемые команды, содержащиеся в оперативной памяти (402).
Память (402), как правило, выполнена в виде ОЗУ и содержит необходимую программную логику, обеспечивающую требуемый функционал.
Средство хранения данных (403) может выполняться в виде HDD, SSD дисков, рейд массива, сетевого хранилища, флэш-памяти, оптических накопителей информации (CD, DVD, MD, Blue-Ray дисков) и т.п. Средство (403) позволяет выполнять долгосрочное хранение различного вида информации.
Интерфейсы (404) представляют собой стандартные средства для подключения и работы с серверной частью, например, USB, RS232, RJ45, LPT, COM, HDMI, PS/2, Lightning, FireWire и т.п.
Выбор интерфейсов (404) зависит от конкретного исполнения устройства (400), которое может представлять собой персональный компьютер, мейнфрейм, серверный кластер, тонкий клиент, смартфон, ноутбук и т.п.
В качестве средств В/В данных (405) в любом воплощении системы должна использоваться клавиатура. Аппаратное исполнение клавиатуры может быть любым известным: это может быть, как встроенная клавиатура, используемая на ноутбуке или нетбуке, так и обособленное устройство, подключенное к настольному компьютеру, серверу или иному компьютерному устройству. Подключение при этом может быть, как проводным, при котором соединительный кабель клавиатуры подключен к порту PS/2 или USB, расположенному на системном блоке настольного компьютера, так и беспроводным, при котором клавиатура осуществляет обмен данными по каналу беспроводной связи, например, радиоканалу, с базовой станцией, которая, в свою очередь, непосредственно подключена к системному блоку, например, к одному из USB-портов. Помимо клавиатуры, в составе средств В/В данных также может использоваться: джойстик, дисплей (сенсорный дисплей), проектор, тачпад, манипулятор мышь, трекбол, световое перо, динамики, микрофон и т.п.
Средства сетевого взаимодействия (406) выбираются из устройств, обеспечивающих сетевой прием и передачу данных, например, Ethernet карту, WLAN/Wi-Fi модуль, Bluetooth модуль, BLE модуль, NFC модуль, IrDa, RFID модуль, GSM модем и т.п. С помощью средств (405) обеспечивается организация обмена данными по проводному или беспроводному каналу передачи данных, например, WAN, PAN, ЛВС (LAN), Интранет, Интернет, WLAN, WMAN или GSM, 3G, 4G, 5G.
Компоненты устройства (400) сопряжены посредством общей шины передачи данных (407).
В настоящих материалах заявки представлено предпочтительное раскрытие осуществления заявленного технического решения, которое не должно использоваться как ограничивающее иные, частные воплощения его реализации, которые не выходят за рамки испрашиваемого объема правовой охраны и являются очевидными для специалистов в соответствующей области техники.
Специалисту в данной области техники должно быть понятно, что различные вариации заявляемого способа и системы не изменяют сущность изобретения, а лишь определяют его конкретные воплощения и применения.
Источники
[1] Emig, D. et al. Drug Target Prediction and Repositioning Using an Integrated Network-Based Approach. PLOS ONE 8, e60618 (2013).
[2] Yue, X. et al. Graph embedding on biomedical networks: methods, applications and evaluations. Bioinformatics btz718 (2019) doi:10.1093/bioinformatics/btz718.
[3] Wang, Z., Zhou, M. & Arnold, C. Toward heterogeneous information fusion: bipartite graph convolutional networks for in silico drug repurposing. Bioinformatics 36, i525–i533 (2020).
[4] https://arxiv.org/pdf/2002.01680.pdf
[5] https://arxiv.org/pdf/2004.00216v3.pdf
[6] http://www.ebi.ac.uk/efo/EFO_0001360
[7] http://www.ebi.ac.uk/efo/EFO_0001073

Изобретение относится к медицине, а именно к терапии, и может быть использовано для предсказания и приоритизации лекарственных мишеней для заболеваний на основе болезнь-специфичного графа с помощью методов машинного обучения. Создают граф белок-белковых взаимодействий, в котором узлами являются белок-кодирующие гены, ребрами являются взаимодействия белок-кодирующих генов. Создают болезнь-специфичный граф на основе графа белок-белковых взаимодействий путем добавления в граф узлов заболеваний и лекарственных мишеней и следующих связей: белок-кодирующий ген - болезнь, белок-кодирующий ген - лекарственная мишень, болезнь - лекарственная мишень, болезнь-болезнь. Разметку узлов и построение связей графа осуществляют с помощью болезнь-специфической информации. С помощью сверточных нейронных сетей осуществляют свертку полученного графа в векторное представление. Подают полученное векторное представление болезнь-специфичного графа в обученную модель машинного обучения для предсказания и приоритизации лекарственных мишеней и получают ранжированный список белок-кодирующих генов с точки зрения вероятности быть лекарственной мишенью для заболеваний. Способ обеспечивает точное прогнозирование вероятности белок-кодирующих генов быть лекарственными мишенями для заболеваний, ускоряет процесс идентификации лекарственных мишеней для заболеваний, и приводит к ускорению разработки новых эффективных лекарств за счет автоматического ранжирования списка белок-кодирующих генов с точки зрения вероятности быть лекарственной мишенью для заболеваний с помощью методов машинного обучения на основе болезнь-специфичного графа. 5 з.п. ф-лы, 5 ил., 5 табл.
1. Компьютерно-реализуемый способ предсказания и приоритизации лекарственных мишеней для заболеваний на основе болезнь-специфичного графа с помощью методов машинного обучения, в котором на вычислительном устройстве, содержащем процессор и память, хранящую исполняемые процессором инструкции, исполняют инструкции, включающие этапы, на которых: создают граф белок-белковых взаимодействий, в котором узлами являются белок-кодирующие гены, ребрами являются взаимодействия белок-кодирующих генов;
создают болезнь-специфичный граф на основе графа белок-белковых взаимодействий путем добавления в граф белок-белковых взаимодействий узлов заболеваний и лекарственных мишеней и следующих связей: белок-кодирующий ген - болезнь, белок-кодирующий ген - лекарственная мишень, болезнь - лекарственная мишень, болезнь-болезнь, причем разметку узлов и построение связей болезнь-специфичного графа осуществляют с помощью болезнь-специфической информации, включающей текстовые и омиксные данные;
с помощью сверточных нейронных сетей осуществляют свертку полученного болезнь-специфичного графа в векторное представление, и для каждого узла/белок-кодирующего гена получают вектор, который характеризует топологические свойства болезнь-специфичного графа и данные дифференциальной экспрессии генов для заболеваний;
подают полученное векторное представление болезнь-специфичного графа в обученную модель машинного обучения для предсказания и приоритизации лекарственных мишеней и на выходе указанной модели получают ранжированный список белок-кодирующих генов с точки зрения вероятности быть лекарственной мишенью для заболеваний.
2. Способ по п. 1, характеризующийся тем, что текстовые и омиксные данные включают, по меньшей мере, следующее: данные о дифференциальной экспрессии генов, данные о связях заболеваний с лекарственными мишенями, данные об онтологии заболеваний, данные о лекарственных мишенях, данные о свойствах белков.
3. Способ по п. 1, характеризующийся тем, что валидируют модель машинного обучения для предсказания и приоритизации лекарственных мишеней при помощи методов обогащения топ К и статистически гипергеометрического теста.
4. Способ по п. 1, характеризующийся тем, что валидируют модель машинного обучения для предсказания и приоритизации лекарственных мишеней по времени.
5. Способ по п. 4, характеризующийся тем, что для валидации модели машинного обучения для предсказания лекарственных мишеней по времени используют обучающий набор, полученный на основе данных, которые были известны до заданной временной точки, и тестовый набор, полученный на основе данных, которые стали известны после указанной заданной временной точки.
6. Способ по п. 5, характеризующийся тем, что используют строгую и нестрогую стратегию для получения обучающего набора, полученного на основе данных, которые были известны до заданной временной точки, и тестового набора, полученного на основе данных, которые стали известны после указанной заданной временной точки, причем при нестрогой стратегии осуществляется деление по заданной временной точке на обучающий и тестовый набор только на основе даты возникновения связей белок-кодирующий ген – болезнь, при строгой стратегии осуществляется деление по заданной временной точке на обучающий и тестовый набор как на основе даты возникновения связей белок-кодирующий ген – болезнь, так и на основе проверки других связей болезнь-специфичного графа.
| US 20190303535 A1, 03.10.2019 | |||
| US 20190164632 A1, 30.05.2019 | |||
| US 20210081717 A1, 18.03.2021 | |||
| US 8332158 B2, 11.12.2012 | |||
| MORDELET F | |||
| et al | |||
| ProDiGe: Prioritization Of Disease Genes with multitask machine learning from positive and unlabeled examples | |||
| BMC Bioinformatics | |||
| Способ приготовления лака | 1924 |
|
SU2011A1 |
| SCHNAUBELT M | |||
| A comparison of machine learning model validation | |||
Авторы
Даты
2023-06-28—Публикация
2022-03-14—Подача