Область техники
[0001] Настоящее изобретение относится к пептиду, который вводят в организм пациента для создания гуморального иммунитета, что представляет собой технологию в области иммунотерапевтических средств.
[0002]
Уровень техники
[0003] Цель иммунотерапевтического средства состоит во введении в организм пациента для индукции гуморального иммунитета против самого иммунотерапевтического средства и, таким образом, для лечения конкретной болезни или заболевания с помощью полученного в результате антитела. В частности, «лечение» также включает предотвращение конкретной болезни или заболевания. Иммунотерапевтические средства аналогичны вакцинам в том, что они индуцируют выработку антител посредством реакции антиген-антитело в организме пациента; однако они отличаются от вакцин тем, что антитело обладает не только способностью связывания с самим иммунотерапевтическим средством, но также способностью связывания с определенной мишенью в организме (например, с конкретными тканями и клетками в организме или с веществами, образующимися в метаболических процессах и т.д.), что позволяет лечить конкретную болезнь или заболевание и осуществлять повторное введение.
[0004]
Описание
Техническая проблема
[0005] Настоящее описание относится к пептиду, который имеет функцию индукции предварительно сконструированного антитела в организме пациента.
[0006] Настоящее описание относится к композиции для иммунотерапевтического средства, включающей пептид.
[0007] Настоящее описание относится к последовательности нуклеиновой кислоты, кодирующей пептид.
[0008] Настоящее описание относится к применению пептида и композиции для иммунотерапевтического средства, включающей пептид.
[0009]
Техническое решение
[0010] В соответствии с аспектом настоящего изобретения предложена пептидная единица (блок пептида) длиной от 23-мера до 71-мера, распознаваемая CD4+Т-клетками для индукции гуморального иммунитета и включающая следующее:
[0011] по меньшей мере один Th-эпитоп, где длина Th-эпитопа составляет от 8-мера до 32-мера; и
[0012] по меньшей мере один В-клеточный эпитоп,
[0013] где В-клеточный эпитоп представляет собой фрагмент или мимотоп аполипопротеина В-100, который может индуцировать антитело, нацеленное на аполипопротеин В-100.
[0014] В одном варианте осуществления пептидная единица имеет длину в диапазоне от 26-мера до 50-мера, а Th-эпитоп имеет длину в диапазоне от 11-мера до 13-мера.
[0015] В одном варианте осуществления пептидная единица включает один В-клеточный эпитоп и один Th-эпитоп, и пептидная единица имеет длину в диапазоне от 26-мера до 45-мера.
[0016] В одном варианте осуществления пептидная единица включает один В-клеточный эпитоп и два Th-эпитопа (каждый из которых упоминается как первый Th-эпитоп и второй Th-эпитоп); пептидная единица имеет длину в диапазоне от 37-мера до 50-мера; и первый Th-эпитоп связан между В-клеточным эпитопом и вторым Th-эпитопом.
[0017] В одном варианте осуществления пептидная единица включает два В-клеточных эпитопа (каждый из которых упоминается как первый В-клеточный эпитоп и второй В-клеточный эпитоп) и один Th-эпитоп; пептидная единица имеет длину в диапазоне от 45-мера до 50-мера; и второй В-клеточный эпитоп связан между первым В-клеточным эпитопом и Th-эпитопом.
[0018] В одном варианте осуществления пептидная единица включает два В-клеточных эпитопа (каждый из которых упоминается как первый В-клеточный эпитоп и второй В-клеточный эпитоп) и один Th-эпитоп; пептидная единица имеет длину в диапазоне от 45-мера до 50-мера; и Th-эпитоп связан между первым В-клеточным эпитопом и вторым В-клеточным эпитопом.
[0019] Настоящее описание относится к нуклеиновой кислоте, кодирующей пептидную единицу или пептид, которые не включают нестандартную аминокислоту для пептидов.
[0020] Настоящее описание относится к пептиду, в котором 2 или более и 5 или менее пептидных единиц связаны между собой.
[0021] Настоящее описание относится к фармацевтической композиции для лечения ожирения, включающей следующее: пептидную единицу или пептид; и адъюванты.
[0022] Настоящее описание относится к способу лечения ожирения, включающему следующее: введение фармацевтической композиции в организм пациента.
[0023] Настоящее описание относится к применению пептидной единицы или пептида для лечения ожирения.
[0024] Настоящее описание относится к применению пептидной единицы или пептида для приготовления терапевтического средства для лечения ожирения.
[0025]
Полезные эффекты изобретения
[0026] Когда пептид, предложенный в настоящем описании, вводится инъекцией в организм пациента, пептид обладает эффектом индукции выработки антитела с определенной физиологической функцией, которое специфически связывается с ранее сконструированным антигенным сайтом.
[0027]
Краткое описание чертежей
[0028] ФИГ. 1-3, которые показывают результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 2, представляют собой графики, показывающие измеренные изменения массы тела в неделю для каждой экспериментальной группы.
[0029] ФИГ. 4, который показывает результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 3, представляет собой график, показывающий измеренную массу на недельный возраст испытуемого для каждой экспериментальной группы, где Lean обозначает контрольную группу с нормальной массой, Obesity обозначает группу ожирения, вызванного диетой с высоким содержанием жиров, Mock обозначает группу, которой вводили плацебо, и 3H-OTP обозначает группу 2-1.
[0030] ФИГ. 5, который показывает результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 3, представляет собой график, показывающий измеренный титр антител, наблюдаемый у испытуемого для каждой экспериментальной группы, в которой Obesity обозначает группу ожирения, вызванного диетой с высоким содержанием жиров, Mock обозначает группу, которой вводят плацебо, а 3H-OTP обозначает группу 2-1.
[0031] ФИГ. 6, который показывает результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 3, представляет собой график, показывающий измеренную способность гормончувствительной липазы к липолизу в адипоцитах испытуемого для каждой экспериментальной группы, где Basal означает случай, когда норэпинефрином не лечили, и Hormone обозначает случай, когда лечили норэпинефрином, а Lean обозначает контрольную группу с нормальной массой, Obesity обозначает группу ожирения, вызванного диетой с высоким содержанием жиров, Mock обозначает группу, получавшую плацебо, а 3H-OTP обозначает группу 2-1 (а) концентрация глицерина, выделяемого на 105 адипоцитов, (b) концентрация глицерина, выделяемого на один грамм адипоцитов.
[0032] ФИГ. 7, который показывает результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 3, представляет собой изображения, показывающие измеренный размер адипоцитов испытуемого для каждой экспериментальной группы, в которой Lean обозначает контрольную группу с нормальной массой, а Obese обозначает группу ожирения, вызванного диетой с высоким содержанием жиров (показаны изображения, окрашенные DAPI, изображения, окрашенные LipidTOX, и совмещенные изображения, соответственно).
[0033] ФИГ. 8, который показывает результаты эксперимента по подтверждению пептидного эффекта в соответствии с экспериментальным примером 3, представляет собой изображения, показывающие измеренный размер адипоцитов испытуемого для каждой экспериментальной группы (показаны окрашенные DAPI изображения ядер и липидов адипоцитов для каждой экспериментальной группы), где Lean обозначает контрольную группу с нормальной массой, Obese обозначает группу ожирения, вызванного диетой с высоким содержанием жиров, Mock обозначает группу, получающую плацебо, а 3H-OTP обозначает группу 2-1.
[0034] ФИГ. 9, который показывает результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 3, представляет собой графики, показывающие измеренную концентрацию липидов в крови испытуемого для каждой экспериментальной группы, в которой TG обозначает триглицерид, NEFA обозначает неэтерифицированную жирную кислоту, CHOL обозначает холестерин, HDL обозначает липопротеин высокой плотности, а LDL обозначает липопротеин низкой плотности.
[0035] ФИГ. 10, который показывает результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 4, представляет собой график, показывающий измеренную массу тела испытуемого в возрасте недели для каждой экспериментальной группы, в которой Lean обозначает контрольную группу с нормальной массой, Obese обозначает группу ожирения, вызванного диетой с высоким содержанием жиров, Mock обозначает группу, получающую плацебо, 3H-OTP 30 мкг обозначает группу 3-2, а 3H-OTP 50 мкг обозначает группу 3-1. Для справки также показан график, относящийся к 3H-OTP-2W 50 мкг, представляющий группу 2-1.
[0036] ФИГ. 11, который показывает результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 4, представляет собой график, показывающий измеренный титр антител испытуемого в возрасте недели для каждой экспериментальной группы, в которой Lean обозначает контрольную группу с нормальной массой, Obese обозначает группу ожирения, вызванного диетой с высоким содержанием жиров, Mock обозначает группу, получающую плацебо, 3H-OTP 30 мкг обозначает группу 3-2, а 3H-OTP 50 мкг обозначает группу 3-1.
[0037] ФИГ. 12, который показывает результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 5, представляет собой график, показывающий измеренную массу тела испытуемого в возрасте недели для каждой экспериментальной группы, в которой Lean обозначает контрольную группу с нормальной массой, Obese обозначает группу ожирения, вызванного диетой с высоким содержанием жиров, а 3H-OTP обозначает группу 4-1.
[0038] ФИГ. 13, который показывает результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 6, представляет собой графики, показывающие увеличение массы тела в возрасте 16 недель по сравнению с таковой в возрасте 11 недель после измерения массы тела испытуемого в возрасте недели для каждой экспериментальной группы по возрасту, где Wild(+/+)-Lean обозначает контрольную группу с нормальной массой, Wild(+/+) обозначает группу 5-1, Hetero(+/-) обозначает группу 5 -2, а Homo(-/-) обозначает группу 5-3.
[0039] ФИГ. 14-22, которые показывают результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 7, и представляют собой графики, показывающие измеренную массу тела испытуемого в возрасте недели в каждой экспериментальной группе, в которой Lean обозначает контрольную группу с нормальной массой, Obese обозначает группу ожирения, вызванного диетой с высоким содержанием жиров, P1 обозначает группу 6-1, P2 обозначает группу 6-2, P3 обозначает группу 6-3, P4 обозначает группу 6-4, P5 обозначает группу 6-5, P6 обозначает группу 6-6, P7 обозначает группу 6-7, P8 обозначает группу 6-8 и P9 обозначает группу 6-9.
[0040] ФИГ. 23-25, которые показывают результаты эксперимента по подтверждению эффекта пептида в соответствии с экспериментальным примером 7, и представляют собой Таблицы, описывающие измеренные титры антител у испытуемого в возрасте 11 недель, 16 недель и 19 недель для каждой экспериментальной группы, в которой Lean обозначает контрольную группу с нормальной массой, Obese обозначает группу ожирения, вызванного диетой с высоким содержанием жиров, P1 обозначает группу 6-1, P2 обозначает группу 6-2, P3 обозначает группу 6-3, P4 обозначает группу 6-4, P5 обозначает группу 6-5, P6 обозначает группу 6-6, P7 обозначает группу 6-7, P8 обозначает группу 6-8 и P9 обозначает группу 6-9. Кроме того, значение, обозначенное меткой в столбце «No» для каждой экспериментальной группы в каждой Таблице, обозначает экспериментальные результаты каждого отдельного пациента в каждой экспериментальной группе, «ave» обозначает общее среднее значение, «sd» обозначает общее стандартное отклонение, а «se» обозначает общую стандартную ошибку.
[0041]
Подробное описание осуществления изобретения
[0042] В дальнейшем раскрытый настоящим объект изобретения будет описан более подробно в терминах некоторых конкретных вариантов осуществления и примеров со ссылкой на прилагаемые чертежи. Следует отметить, что прилагаемые чертежи охватывают некоторые, но не все варианты осуществления настоящего описания. Раскрываемый настоящим объект изобретения может быть реализован во многих различных формах и не должен рассматриваться как ограниченный конкретными вариантами осуществления, изложенными в настоящем документе. Скорее, эти варианты осуществления предложены для того, чтобы это описание удовлетворяло применимым юридическим требованиям. Действительно, специалистам в данной области техники будет понятно множество модификаций и других вариантов осуществления настоящего описания, к которым относится раскрытый настоящим объект изобретения. Следовательно, следует понимать, что раскрытый настоящим объект изобретения не должен ограничиваться конкретными раскрытыми вариантами осуществления и что модификации и другие варианты осуществления предназначены для включения в объем прилагаемой формулы изобретения.
[0043]
[0044] Определения общих терминов
[0045] примерно
[0046] Используемый в настоящем описании термин «примерно» относится к степени, близкой к определенному количеству, и относится к количеству, уровню, значению, числу, частоте, проценту, измерению, размеру, количеству, массе или длине, которые варьируются в пределах 30%, 25%, 20%, 25%, 10%, 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2% или 1% по отношению к эталонному количеству, уровню, значению, числу, частоте, проценту, измерению, размеру, количеству, весу или длине.
[0047] пептид
[0048] Используемый в настоящем описании термин «пептид» относится к полимеру аминокислот. Термин «пептид» относится к форме, в которой связано небольшое количество аминокислот, и в основном используется для того, чтобы отличить его от белка. Не существует четкого стандарта для отличия белков от пептидов, но, при использовании в настоящем документе, в то время как полимеры из примерно 200 аминокислот называются пептидами, те, что больше, называются белками, если не указано иное. Термин «пептид» может включать все другие значения, известные специалистам в данной области.
[0049] субъект
[0050] Используемый в настоящем описании термин «субъект» относится к организму, который представляет собой субъект, подвергшийся воздействию определенного вещества (например, пептида и т.д.). Субъект может относиться к независимому организму (например, человеку, животному и т.д.) или может относиться к неполной конструкции независимого организма (например, части ткани, клетки и т.д.). Это значение может быть правильно истолковано в зависимости от контекста. Кроме того, термин «субъект» может дополнительно включать все значения, признанные специалистами в данной области.
[0051] иммунотерапевтические средства
[0052] Используемый в настоящем описании термин «иммунотерапевтические средства» представляет собой понятие, отличное от общих терапевтических средств или вакцин. Иммунотерапевтические средства аналогичны существующим вакцинам в том смысле, что их вводят в организм пациента для индукции гуморального иммунного ответа против самих иммунотерапевтических средств. Однако иммунотерапевтические средства отличаются от существующих вакцин тем, что антитела, индуцируемые в результате гуморального иммунного ответа, обладают способностью связываться не только с самими иммунотерапевтическими средствами, но и со специфическими тканями и клетками организма (например, рецепторы на клеточной поверхности) или специфическими веществами (например, пептиды, липиды, белки и/или сахариды), образующимися в процессе метаболизма; тем самым они могут подвергать лечению конкретную болезнь или заболевание, и их можно вводить непрерывно и многократно. Соответственно, иммунотерапевтические средства обычно включают антигены, предназначенные для индукции антител, обладающих способностью связываться с определенной тканью-мишенью, клеткой или веществом в организме. Если не указано иное, термин «иммунотерапевтические средства» интерпретируется как включающий все антигены, которые могут быть надлежащим образом использованы специалистами в данной области (например, пептиды, белки, липиды, сахариды и/или их комплексы и т.д.), обладающие вышеописанными функциями. Термин «иммунотерапевтические средства» может быть более ограниченно обозначен как «гуморально-иммунотерапевтические средства». Кроме того, термин «иммунотерапевтические средства» может включать все значения, признанные специалистами в данной области.
[0053] лечение или терапия
[0054] Используемый в настоящем описании термин «лечение» в совокупности относится к любому прямому или косвенному действию или мере, направленной на устранение, облегчение, уменьшение, ингибирование или улучшение заболевания, болезни, расстройства и/или симптомов пациента, или к любому прямому или косвенному действию или мере, направленным на получение результатов предотвращения заболевания, болезни, расстройства и/или симптомов. Используемый в настоящем описании термин «терапевтические средства» относится к различным веществам (например, соединениям или пептидам), которые могут проявлять «лечебный» эффект при соответствующем введении пациенту. Кроме того, термин «лечение» или «терапевтические средства» может включать все другие значения, признанные специалистами в данной области.
[0055] иммуногенность
[0056] Используемый в настоящем описании термин «иммуногенность» в совокупности относится к «свойству действовать как антиген, способный индуцировать иммунный ответ» согласно определению. Существуют различные методы измерения иммуногенности конкретного антигена, и эти методы могут быть соответствующим образом адаптированы или разработаны в соответствии с целью. Например, способы могут включать 1) способ подтверждения образования антител типа IgG, IgA и/или IgE в организме пациента при введении антигена в организм пациента, 2) способ подтверждения времени образования антител типа IgG, IgA и/или IgE в зависимости от цикла введения, 3) способ подтверждения титра индуцированных антител к антигену и 4) способ измерения эффекта по механизму действия, когда обнаружен механизм действия индуцированных антител, но способы этим не ограничиваются. Выражение «повышение иммуногенности» может использоваться взаимозаменяемо, например, с «усилением эффекта индукции иммунного ответа», «улучшением способности индуцировать антитела» и «повышением эффективности в качестве иммунотерапевтического средства», и он включает все выражения, которые специалисты в данной области техники могут правильно интерпретировать в соответствии с контекстом.
[0057] «-мер»
[0058] Используемый в настоящем описании термин «-мер» обычно относится к количеству звеньев в полимере с высокой молекулярной массой. Используемый в настоящем описании термин «-мер» обычно выражается как «пептид длиной, соответствующей N-меру» вместе с числом при выражении длины пептида, который относится к пептиду, в котором полимеризовано N аминокислот. Единица, обозначенная выражением «-мер», должна правильно интерпретироваться в контексте, и она включает все другие значения, которые могут быть распознаны специалистами в данной области.
[0059] стандартная аминокислота
[0060] Используемый в настоящем описании термин «стандартная аминокислота» относится к 20 аминокислотам, синтезируемым посредством процессов транскрипции и трансляции генов в организме организма. В частности, стандартная аминокислота включает аланин (Ala, A), аргинин (Arg, R), аспарагин (Asn, N), аспарагиновую кислоту (Asp, D), цистеин (Cys, C), глутаминовую кислоту (Glu, E), глутамин (Gln, Q), глицин (Gly, G), гистидин (His, H), изолейцин (Ile, I), лейцин (Leu, L), лизин (Lys, K), метионин (Met, M), фенилаланин (Phe, F), пролин (Pro, P), серин (Ser, S), треонин (Thr, T), триптофан (Trp, W), тирозин (Tyr, Y) и валин (Val, V). Стандартная аминокислота имеет соответствующий кодон ДНК и может быть представлена обычным однобуквенным или трехбуквенным обозначением аминокислоты. Объекты, упоминаемые термином «стандартная аминокислота», должны интерпретироваться соответствующим образом в соответствии с контекстом, и они включают все другие значения, которые могут быть распознаны специалистами в данной области.
[0061] нестандартная аминокислота
[0062] Используемый в настоящем описании термин «нестандартная аминокислота» относится к аминокислоте, отличной от стандартной аминокислоты. К нестандартной аминокислоте относятся искусственные и неприродные аминокислоты, а также те аминокислоты, которые химически модифицированы путем посттрансляционной модификации внутри организма и т.д. К нестандартной аминокислоте относятся, например, D-форма аланина, L-циклогексилаланин, 6-аминогексановая кислота и др. Поскольку нестандартная аминокислота не имеет соответствующего кодона ДНК, она не может быть представлена общим однобуквенным или трехбуквенным обозначением аминокислоты, а записывается другими символами и поясняется дополнительным пояснением. Объекты, обозначаемые термином нестандартной аминокислоты, должны интерпретироваться соответствующим образом в соответствии с контекстом, и они включают все другие значения, которые могут быть распознаны специалистами в данной области.
[0063] описание пептидной последовательности
[0064] Если не указано иное, при описании последовательности пептида в настоящем описании используется однобуквенное обозначение или трехбуквенное обозначение аминокислоты, и оно записывается в направлении от N-конца к С-концу. Например, когда пептид представлен как RNVP, то он относится к пептиду, в котором аргинин, аспарагин, валин и пролин последовательно связаны в направлении от N-конца к С-концу. В другом примере, когда пептид выражен как Thr-Leu-Lys, то он относится к пептиду, в котором треонин, лейцин и лизин последовательно связаны в направлении от N-конца к С-концу. В случае аминокислот, которые не могут быть представлены однобуквенным обозначением, для описания этих аминокислот используются другие буквы, которые будут объяснены в дополнительных пояснениях.
[0065] При выражении пептида в виде структурной формулы N- и -C могут использоваться для четкого обозначения N-конца или C-конца и могут быть подчеркнуты, чтобы различать N-конец и/или C-конец. Например, когда структурная формула пептида выражается как N-B-T-A-C, «N-», написанное в начале, и «-C», написанное в конце, являются символами, указывающими на направления N-конца и С-конца, если не указано иное. Это относится к пептиду, в котором последовательности, представленные B, T и A, связаны в направлении от N-конца к C-концу.
[0066] УРОВЕНЬ ТЕХНИКИ - ГУМОРАЛЬНЫЙ ИММУНИТЕТ
[0067] гуморальный иммунитет с помощью иммуноглобулина М (IgM)
[0068] Среди гуморальных иммунитетов IgM-индуцированный иммунный ответ представляет собой врожденную иммунную функцию, которая в основном активна при первичном иммунном ответе и быстро возникает на ранних стадиях инфекции. IgM в основном секретируется в форме пентамера и теоретически имеет 10 антигенсвязывающих сайтов, поэтому он может одновременно связываться с большим количеством антигенов. Хотя IgM может связываться с широким спектром типов и форм антигенов, аффинность и авидность связывания ограничиваются внутренней аффинностью самого IgM. Следовательно, аффинность и авидность IgM к антигену значительно ниже, чем у антитела, такого как IgG, генерируемого с помощью хелперных Т-клеток.
[0069] ограничения гуморального иммунитета IgM
[0070] Хотя IgM-индуцированный гуморальный иммунитет играет важную роль в начальном иммунном ответе, эффект на основе гуморального иммунитета IgM ограничен, поскольку 1) продукция IgM, продуцируемого В-клетками, низка по сравнению с другими типами антител (например, IgG, продуцируемые дифференцированными В-клетками и др.), 2) специфическая связывающая способность IgM к антигену низкая по сравнению с IgG и 3) степень вторичного иммунного ответа слабая при повторном воздействии того же антигена. Следовательно, с точки зрения конструирования антигена, индуцирующего иммунный ответ, в случае, когда антиген, введенный в организм пациента, индуцирует только гуморальный иммунитет посредством IgM, весьма вероятно, что желаемый эффект не будет получен. Следовательно, очень важно сконструировать антиген так, чтобы IgG индуцировал гуморальный иммунитет.
[0071] гуморальный иммунитет за счет иммуноглобулина G (IgG) 1 - обзор
[0072] Гуморальный иммунитет, который производит IgG, в основном возникает в зародышевом центре селезенки или лимфатического узла и протекает за счет комплексного действия В-клеток, хелперных Т-клеток и антигенпрезентирующих клеток (АПК). Общий процесс выглядит следующим образом. 1) В-клетки распознают вторгшиеся антигены (в основном белковые или пептидные антигены). 2) После того, как антигенпрезентирующие клетки подвергают эндоцитозу антигены (или их фрагменты) и разрезают их на более мелкие фрагменты внутри клеток, а также презентируют некоторые из фрагментов МНС класса II на поверхности антигенпрезентирующих клеток. 3) Хелперные Т-клетки распознают фрагменты антигена, презентированные молекулам MHC класса II. 4) Хелперные Т-клетки передают сигнал дифференцировки В-клеткам (антиген-распознающим клеткам). 5) В-клетки активируются, и некоторые из них дифференцируются в плазматические клетки с образованием IgG-антитела, обладающего высокой специфической способностью связываться с антигенами. 6) В результате активации В-клеток некоторые клетки дифференцируются в В-клетки памяти и сохраняются в организме, чтобы вызвать иммунный ответ, который быстро вырабатывает антиген IgG при повторном вторжении тех же антигенов.
[0073] гуморальный иммунитет за счет IgG 2 - антиген-распознающие клетки
[0074] Антигенпрезентирующие клетки представляют собой собирательный термин для клеток, которые способны к эндоцитозу белковых фрагментов или пептидов, расщеплению их на более короткие пептидные фрагменты, размещении их на молекулах МНС класса II и презентации их на поверхности антигенпрезентирующих клеток. Основные антигенпрезентирующие клетки включают В-клетки, макрофаги, дендритные клетки и т.д. Антигенпрезентирующие клетки транспортируют эндоцитированные фрагменты антигена из места инфекции в лимфатический узел и презентируют фрагменты антигена хелперным Т-клеткам с помощью МНС класса II и тем самым играют роль в индукции иммунного ответа путем активации хелперных Т-клеток, которые распознают их.
[0075] гуморальный иммунитет за счет IgG 3 - MHC класса II
[0076] МНС класса II представляет собой молекулу, которая экспрессируется на поверхности антигенпрезентирующих клеток и имеет гетеродимерную структуру, состоящую из цепей α/β. МНС класса II благодаря своей структуре может связываться с пептидом определенной длины и презентировать его. Антигенпрезентирующие клетки позволяют пептидным фрагментам, полученным из чужеродных антигенов, связываться с МНС класса II и презентировать их на клеточной поверхности. Комплексы генов HLA (комплексы генов лейкоцитарного антигена человека) участвуют в экспрессии MHC класса II у человека, и известно, что среди них такие комплексы генов, как HLA-DP, DQ, DR и т.д., участвуют в экспрессии рецепторов клеточной поверхности MHC II класса на поверхности антигенпрезентирующих клеток. Известно, что у людей ген HLA-DR имеет различные аллели в зависимости от расы, и примерно 12 типов генов HLA-DR известны как наиболее часто встречающиеся аллели.
[0077] гуморальный иммунитет за счет IgG 4 - презентация MHC класса II антигенпрезентирующими клетками
[0078] Хотя между литературными данными имеется небольшая разница, известно, что длина пептида, презентированного в МНС класса II, находится в диапазоне от примерно 17-мера до примерно 24-мера. Следовательно, антигенпрезентирующие клетки не презентируют эндоцитированные антигенные белки или пептидные фрагменты на МНС класса II в том виде, в каком они есть, но они подвергаются процессу расщепления, превращая их в более мелкие фрагменты от 17-мера до 24-мера. Антигенные фрагменты (белки или пептидные фрагменты), эндоцитированные антигенпрезентирующими клетками, находятся в эндосоме, а эндосома слита с лизосомой антигенпрезентирующих клеток. После этого фрагменты антигена расщепляются на более короткие пептиды с помощью различных ферментов деградации, присутствующих в лизосомах. Примеры разрушающих ферментов включают эндопептидазы и экзопептидазы. В то время как эндопептидазы расщепляют фрагменты антигена, воздействуя на пептидную связь внутри фрагментов антигена, экзопептидазы в основном расщепляют фрагменты антигена, воздействуя на пептидные связи на обоих концах фрагментов антигена. Когда фрагменты антигена расщепляются на пептиды подходящего размера с помощью описанного выше процесса, некоторые из них связываются с МНС класса II, присутствующим во внутренней мембране лизосомы. Лизосома возвращается на клеточную поверхность и сливается с клеточной плазматической мембраной, в результате чего МНС класса II и связанные с ним пептидные фрагменты экспонируются на поверхности антигенпрезентирующих клеток. Весь этот процесс также называют «процессом, посредством которого антигенпрезентирующие клетки презентируют антигены».
[0079] гуморальный иммунитет за счет IgG 5 - хелперные Т-клетки
[0080] Хелперные Т-лимфоциты также известны как CD4+клетки, поскольку они экспрессируют CD4. Хелперные Т-клетки экспрессируют Т-клеточные рецепторы (TCR), которые обладают способностью связываться с МНС класса II на поверхности. Рецепторы Т-клеток обычно образуют комплекс с CD3. Когда антигены (например, пептидные фрагменты), транспортируемые антигенпрезентирующими клетками в лимфатические узлы, презентируются через МНС класса II, хелперные Т-клетки распознают фрагменты антигена, представленные выше. В этом процессе распознавания участвуют комплекс Т-клеточный рецептор-CD3 и CD4. Когда хелперные Т-клетки успешно распознают фрагменты антигена, они активируются для секреции различных цитокинов или дифференцировки. Секретируемые цитокины участвуют в дифференцировке В-клеток, которая будет описана позже.
[0081] гуморальный иммунитет за счет IgG 6 - дифференцировка В-клеток
[0082] Под влиянием цитокинов (например, интерлейкина-4 (IL-4) и т.д.), секретируемых хелперными Т-клетками, происходит переключение класса иммуноглобулинов В-клеток, что приводит к изменению изотипов антител, продуцируемых В-клетками. (например, с IgM на IgG). Кроме того, некоторые В-клетки дифференцируются в В-клетки памяти и сохраняются, чтобы вызвать быстрый иммунный ответ при повторном вторжении тех же антигенов, а некоторые дифференцируются в плазматические клетки и активно продуцируют антитела IgG.
[0083] гуморальный иммунитет за счет IgG 7 - условия для продукции IgG
[0084] Для возникновения гуморального иммунитета за счет IgG важно, чтобы 1) специфическая трехмерная структура антигена распознавалась В-клетками и 2) некоторые фрагменты антигена распознавались хелперными Т-клетками через МНС класса II. В целом, хотя часть антигена, распознаваемая В-клетками, и часть антигена, распознаваемая хелперными Т-клетками, отличаются друг от друга и активируют иммунный ответ разными путями, общеизвестно, что иммунный ответ возникает только тогда, когда часть, распознаваемая В-клетками (В-клеточный эпитоп), и часть, распознаваемая хелперными Т-клетками (Th-эпитоп), имеют по меньшей мере определенную связь. Например, В-клеточный эпитоп и Th-эпитоп могут быть включены в одну молекулу, образовывать конъюгат или иметь другие связи.
[0085] ОГРАНИЧЕНИЯ ПРЕДШЕСТВУЮЩЕГО УРОВНЯ ТЕХНИКИ
[0086] что следует учитывать при конструкции пептидов, которые можно использовать в качестве иммунотерапевтических средств
[0087] Как описано выше, иммунотерапевтические средства необходимы благодаря их характеристикам: 1) они способны стабильно индуцировать иммунный ответ в организме пациента, 2) они способны минимизировать побочные эффекты, равномерно индуцируя только заданное антитело в организме пациента; и 3) для их коммерциализации они легко синтезируются, а стоимость их производства является разумной. Таким образом, при конструкции пептида, который можно использовать в качестве иммунотерапевтического средства, необходимо учитывать следующие три условия: 1) пептид должен проявлять определенный уровень иммуногенности, 2) в организме пациента пептид должен вызывать иммунный ответ, который равномерно индуцирует специфичность распознавания антигена антителом, заданным заранее, изотипа, который контролирует физиологические функции антитела, и т.д., и 3) пептид должен быть легко синтезируемым с учетом экономической целесообразности.
[0088] ограничения предшествующего уровня техники
[0089] Как раскрыто в ранее поданных патентных заявках US10/378707 и PCT/KR2005/000784, и Kim et al. (2016, An apolipoprotein B100 mimotope prevents obesity in mice, Clinical Science 130, 105-116), известно, что антитела, специфичные к искусственно полученному пептиду с определенной последовательностью, также могут связываться с экспонированным сайтом белка ApoB-100 в молекуле LDL, и, таким образом, он может функционировать как иммунотерапевтическое средство. Используя такую характеристику, иммунотерапевтические средства, включающие пептид, были разработаны и раскрыты в вышеупомянутых заявках на патенты и т.д. Однако предшествующий уровень техники был в основном направлен на повышение иммуногенности пептида, например, 1) получение длинной непрерывной идентичной последовательности (конкатемер) пептида, 2) конструирование иммунотерапевтического средства путем связывания хелперного Т-клеточного эпитопа (достаточно длинного на уровне белка) с конкатемером и т.д. Соответственно, иммунотерапевтические средства, разработанные традиционным способом, имели ограничения в том, что 1) различные типы антител индуцировались, а однородность снижалась из-за присутствия различных эпитопов (антигенных детерминант), и 2) экономическая целесообразность была низкой из-за высокой себестоимости их производства.
[0090] необходимость создания метода пептидного конструирования
[0091] Что касается пептидов для использования в качестве иммунотерапевтических средств, не было установлено никакого принципа в отношении 1) равномерной индукции только заданного иммунного ответа и 2) конструирования пептида, который легко синтезируется и имеет низкую стоимость производства. Соответственно, в настоящем описании будут представлены технические вопросы, которые необходимо учитывать при конструировании пептидов для применения в качестве иммунотерапевтических средств, и способы их конструирования.
[0092] пептид
[0093] обзор пептидов
[0094] Предложенные в настоящем описании пептиды включают по меньшей мере одну пептидную единицу (блок пептида). Пептидная единица включает по меньшей мере один В-клеточный эпитоп, по меньшей мере один Th-эпитоп и соответствующее количество вспомогательных частей. В одном варианте осуществления пептид может включать одну пептидную единицу. В другом варианте осуществления пептид может включать две или более пептидных единицы.
[0095] характеристика пептида 1 - включение пептидной единицы
[0096] Пептидная единица представляет собой часть, предназначенную 1) для проявления иммуногенности выше определенного уровня и 2) для равномерной индукции только заданного антитела. Таким образом, предложенная в настоящем описании пептидная единица обладает свойствами, подходящими для использования в качестве иммунотерапевтического средства.
[0097] характеристика пептида 2 - относительно короткая длина пептидной единицы
[0098] Поскольку пептидная единица сконструирована с относительно короткой длиной, ее легко синтезировать, а себестоимость производства низкая. Пептид сконструирован с использованием пептидной единицы в качестве его компонента, и, в частности, он разработан в форме, в которой одна или более пептидных единиц связаны. Когда пептид включает только небольшое количество пептидных единиц, общая длина пептида мала, что дает преимущество в простоте синтеза. Даже когда пептид имеет относительно длинную последовательность, включающую множество пептидных единиц, сама пептидная единица хорошо сконструирована для легкого синтеза, и, таким образом, можно получить пептид, синтезируя параллельно пептидные единицы, а затем связывая эти пептидные единицы. В результате пептид по настоящему изобретению характеризуется простотой синтеза, что является характеристикой, подходящей для применения пептида в качестве иммунотерапевтического средства, в дополнение к характеристикам пептидной единицы, описанным выше.
[0099] функции пептида
[00100] Когда пептид вводят в организм пациента, он выполняет функцию равномерной индукции только антител, способных специфически связываться с В-клеточным эпитопом, включенным в пептид.
[00101] В-КЛЕТОЧНЫЙ ЭПИТОП
[00102] определение В-клеточного эпитопа
[00103] Предложенные в настоящем описании пептиды включают один или более В-клеточных эпитопов. Используемый в настоящем описании термин В-клеточный эпитоп относится к единице пептида, предназначенной для индукции гомогенного антитела одного типа. Следовательно, когда пептид, включающий В-клеточный эпитоп, вводят в организм пациента, это приводит к тому, что один тип антител индуцируется доминантно для каждого типа В-клеточного эпитопа.
[00104] структура В-клеточного эпитопа
[00105] В-клеточный эпитоп включает часть для формирования трехмерной структуры и прилегающую к ней часть. Часть для формирования трехмерной структуры представляет собой часть, которая образует пептид со структурой более высокого порядка, и эта часть сконструирована таким образом, чтобы В-клетки могли распознавать пептид со структурой более высокого порядка и продуцировать антитело, которое может специфически связываться с этим пептидом. Смежная часть представляет собой часть, которая прямо или косвенно влияет на часть для формирования трехмерной структуры, чтобы стабильно формировать структуру более высокого порядка. В частности, смежная часть может иметь такие функции, как 1) функция части для формирования трехмерной структуры для формирования конкретной структуры, 2) функция линкера, которая не влияет на часть для формирования трехмерной структуры, когда В-клеточный эпитоп связан с другой частью внутри пептидной единицы, 3) функция защиты части для формирования трехмерной структуры и т.д., но их функции этим не ограничиваются. В одном варианте осуществления В-клеточный эпитоп может иметь последовательность, в которой первая часть для формирования трехмерной структуры и ее первая смежная часть связаны в порядке от N-конца к С-концу. В другом варианте осуществления В-клеточный эпитоп может иметь последовательность, в которой вторая смежная часть, вторая часть для формирования трехмерной структуры и третья смежная часть последовательно связаны в порядке от N-конца к С-концу. Еще в одном варианте осуществления В-клеточный эпитоп может иметь последовательность, в которой третья часть для формирования трехмерной структуры и четвертая смежная часть последовательно связаны в порядке от N-конца к С-концу.
[00106] конструирование В-клеточного эпитопа 1 - конструирование части для формирования трехмерной структуры
[00107] В-клеточный эпитоп должен быть способен равномерно индуцировать продукцию антител, способных распознавать трехмерную структуру В-клеток и специфически связываться с ними. Трехмерная структура, распознаваемая В-клетками, может быть выражена через соответствующий пептид со структурой более высокого порядка. Следовательно, В-клеточный эпитоп сконструирован для включения части для формирования трехмерной структуры, которая образует пептид со структурой более высокого порядка. Часть для формирования трехмерной структуры может образовывать заданный пептид со структурой более высокого порядка в зависимости от назначения. В одном варианте осуществления часть для формирования трехмерной структуры может включать структуру α-спирали. В другом варианте осуществления часть для формирования трехмерной структуры может включать в себя β-структуру. Еще в одном варианте осуществления часть для формирования трехмерной структуры может включать α-спираль и/или β-структуры. В другом варианте часть для формирования трехмерной структуры может включать пептид с третичной структурой. Еще в одном варианте осуществления часть для формирования трехмерной структуры может включать пептид с четвертичной структурой.
[00108] конструирование В-клеточного эпитопа 2 - смежная часть
[00109] При конструировании В-клеточного эпитопа не обязательно, чтобы все последовательности образовывали пептид со структурой более высокого порядка. Другими словами, В-клеточный эпитоп может быть сконструирован так, чтобы дополнительно включать смежную часть, в дополнение к части для формирования трехмерной структуры. Смежная часть может воздействовать на часть для формирования трехмерной структуры, так что она устойчиво формирует структуру более высокого порядка. Смежная часть может выполнять различные другие функции, и ее роль может совпадать с ролью вспомогательной части. В одном варианте осуществления смежная часть может выполнять линкерную функцию. В другом варианте осуществления смежная часть может иметь защитную функцию для части для формирования трехмерной структуры. Еще в одном варианте осуществления смежная часть может выполнять одну или более функций.
[00110] длина В-клеточного эпитопа
[00111] В-клеточный эпитоп должен иметь 1) размер, достаточный для распознавания В-клетками, и 2) один или очень мало типов антител, которые специфически связываются с В-клеточным эпитопом. Длина В-клеточного эпитопа должна быть ограничена до соответствующего уровня. Когда длина В-клеточного эпитопа слишком мала, он не распознается В-клетками и, таким образом, не обладает способностью индуцировать антитела, тогда как, когда длина В-клеточного эпитопа слишком велика, могут индуцироваться различные типы антител, которые отклоняются от заданной цели. В одном варианте осуществления длина В-клеточного эпитопа может соответствовать примерно 8-меру, примерно 9-меру, примерно 10-меру, примерно 11-меру, примерно 12-меру, примерно 13-меру, примерно 14-меру, примерно 15-меру, примерно 16-меру, примерно 17-меру, примерно 18-меру, примерно 20-меру, примерно 21-меру, примерно 22-меру, примерно 23-меру, примерно 24-меру, примерно 25-меру, примерно 26-меру, примерно 27-меру, примерно 28-меру, примерно 29-меру или примерно 30-меру.
[00112] В другом варианте осуществления длина В-клеточного эпитопа может иметь значение в пределах двух числовых диапазонов, выбранных в непосредственно предшествующем предложении.
[00113] варианты В-клеточного эпитопа
[00114] В одном варианте осуществления В-клеточный эпитоп может быть таким, который индуцирует антитело, нацеленное на аполипопротеин В-100. В другом варианте осуществления В-клеточный эпитоп может представлять собой фрагмент аполипопротеина В-100 и/или мимотоп аполипопротеина В-100. Еще в одном варианте осуществления В-клеточный эпитоп отличается тем, что он индуцирует антитело, нацеленное на сайт, выбранный из следующего: открытый сайт аполипопротеина B-100, включенный в липопротеин низкой плотности (LDL); и открытый сайт аполипопротеина B-100, включенный в липопротеин сверхнизкой плотности (VLDL).
[00115] варианты последовательностей В-клеточного эпитопа
[00116] В одном варианте осуществления В-клеточный эпитоп представляет собой пептид, включающий последовательность, выбранную из группы, состоящей из RNVPPIFNDVYWIAF(SEQ ID NO: 6), CRFRGLISLSQVYLS(SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH(SEQ ID NO: 8), RNVPPIFNDVY(SEQ ID NO: 9), CRFRGLISLSQ(SEQ ID NO: 10), KTTKQSFDLSVK(SEQ ID NO: 11), RNVPPIFNDVYW(SEQ ID NO: 12), CRFRGLISLSQV(SEQ ID NO: 13), KTTKQSFDLSVKAQYKK(SEQ ID NO: 14), RNVPPIFNDVYWI(SEQ ID NO: 15), CRFRGLISLSQVY(SEQ ID NO: 16), KTTKQSFDLSVKAQYKKN(SEQ ID NO: 17), PIFNDVYWIAF(SEQ ID NO: 18), GLISLSQVYLS(SEQ ID NO: 19), QSFDLSVKAQYKKNKH(SEQ ID NO: 20), PPIFNDVYWIAF(SEQ ID NO: 21), RGLISLSQVYLS(SEQ ID NO: 22), KQSFDLSVKAQYKKNKH(SEQ ID NO: 23), VPPIFNDVYWIAF(SEQ ID NO: 24), FRGLISLSQVYLS(SEQ ID NO: 25), TKQSFDLSVKAQYKKNKH(SEQ ID NO: 26), NVPPIFNDVYWIA(SEQ ID NO: 27), RFRGLISLSQVYL(SEQ ID NO: 28), TKQSFDLSVKAQYKKN(SEQ ID NO: 29), VPPIFNDVYWI(SEQ ID NO: 30), FRGLISLSQVY(SEQ ID NO: 31), TKQSFDLSVKAQYKKN(SEQ ID NO: 32), PPIFNDVYW(SEQ ID NO: 33), RGLISLSQV(SEQ ID NO: 34), KQSFDLSVKAQYKK(SEQ ID NO: 35), RFRGLISLSQVYLDP(SEQ ID NO: 221), и SVCGCPVGHHDVVGL(SEQ ID NO: 222).
[00117] В другом варианте осуществления В-клеточный эпитоп может представлять собой пептид, который включает эпитоп, включенный в пептид, выбранный из группы, состоящей из SEQ ID NO: 6-35 и 221-222.
[00118] последовательности, сходные с типичными последовательностями В-клеточного эпитопа
[00119] В настоящем описании раскрыты последовательности, сходные с типичными последовательностями В-клеточного эпитопа. В одном варианте осуществления В-клеточный эпитоп может иметь последовательность, имеющую 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с теми последовательностями, которые выбраны из группы, состоящей из SEQ ID NO: 6-34 и SEQ ID NO: 221-222. В другом варианте осуществления В-клеточный эпитоп может представлять собой последовательность, которая соответствует выбранной последовательности более чем на значение, выбранное в непосредственно предшествующем предложении. Например, В-клеточный эпитоп может иметь последовательность, которая на 90% или более идентична SEQ ID NO: 6.
[00120] Th ЭПИТОП
[00121] определение Th-эпитопа
[00122] Th-эпитоп, включенный в предложенный в настоящем описании пептид, относится к части, которая сконструирована таким образом, что после эндоцитоза пептида антигенпрезентирующими клетками он связывается с МНС класса II, презентируется на поверхности антигенпрезентирующей клетки и функционирует для распознавания хелперными Т-клетками (Th, хелперные-Т-лимфоциты), в процессе презентации на поверхности антигенпрезентирующих клеток МНС класса II. Ранее был описан процесс презентации антигенов, при котором антигенпрезентирующие клетки обрабатывают эндоцитированный пептид и позволяют ему связываться с МНС класса II. Другими словами, Th-эпитоп представляет собой часть, которая играет роль в распознавании хелперными Т-клетками, когда пептид вводят инъекцией в организм пациента; следовательно, он играет непосредственную роль в индукции антител типа IgG против пептида.
[00123] конструирование Th-эпитопа 1 - якорный остаток
[00124] Th-эпитоп сконструирован таким образом, что в его последовательности имеется якорный остаток, способный связываться с МНС класса II. Включение якорного остатка в последовательность является важным фактором, влияющим на функцию Th-эпитопа. В одном варианте осуществления Th-эпитоп может включать в качестве якорных остатков одну или более аминокислот, выбранных из группы, состоящей из тирозина (Y), фенилаланина (F), триптофана (W), аргинина (R), лейцина (L), валина (V), изолейцина (I) и метионина (M).
[00125] конструкция Th-эпитопа 2 - видоспецифичный Th-эпитоп
[00126] В качестве Th-эпитопа может быть выбран Th-эпитоп, обладающий способностью связываться с МНС класса II определенного вида, в соответствии с его назначением. В одном варианте осуществления Th-эпитоп может представлять собой Th-эпитоп, обладающий способностью связываться с MHC класса II человека. В другом варианте осуществления Th-эпитоп может представлять собой Th-эпитоп, обладающий способностью связываться с МНС класса II вида, принадлежащего млекопитающим. В частности, Th-эпитоп может представлять собой Th-эпитоп, обладающий способностью связываться с МНС класса II мыши.
[00127] конструирование Th-эпитопа 3 - ген-специфический Th-эпитоп
[00128] Из-за разнообразных признаков комплекса генов HLA структура MHC класса II может различаться у представителей разных рас и индивидуумов. Соответственно, можно сконструировать Th-эпитоп, обладающий способностью связываться с HLA-DP, HLA-DQ и/или HLA-DR, которые представляют собой молекулы МНС класса II с определенным генетическим признаком. В одном варианте осуществления Th-эпитоп может представлять собой пептидную последовательность, обладающую высокой способностью связывания с молекулами MHC класса II, экспрессируемыми одним или более генами HLA-DR, выбранными из 2w2b, 2w2a, 3, 4w4, 4w14, 5, 7, 52a, 52b, 52c и 53, которые представляют собой аллели HLA-DR1.
[00129] В одном варианте осуществления Th-эпитоп может представлять собой пептидную последовательность, обладающую высокой способностью связывания с молекулами MHC класса II, экспрессируемыми одним или более генами, выбранными из HLA-DQ5, HLA-DR, HLA-DR1, HLA-DR8, HLA-DR11, HLA-DR13, HLA-DR14, HLA-DRw52, HLA-DR2w15, HLA-DPw4, каждого подтипа HLA-DRB1 (например, 0301, 01, 03, 04, 07, 08, 09, 11, 12, 13, 15 и 0301) и HLA-DRB5.
[00130] В другом варианте осуществления Th-эпитоп может представлять собой последовательность, названную НА307-312, описанную у Cara C. Wilson et al. (2001, Identification and Antigenicity of Broadly Cross-Reactive and Conserved Human Immunodeficiency Virus Type 1-Derived Helper T-Lymphocyte Epitopes, Journal of Virology, 75(9) 4195-4207).
[00131] Еще в одном варианте осуществления Th-эпитоп может представлять собой один из рестриктированных эпитопов HLA класса II, раскрытых в Таблице 2 работы Christopher P Desmond et al. (2008, A systematic review of T-cell epitopes in hepatitis B virus: identification, genotypic variation and relevance to antiviral therapeutics, Antiviral Therapy 13:161-175).
[00132] конструирование Th-эпитопа 4 - ген-неспецифический Th-эпитоп
[00133] Независимо от признаков комплекса генов HLA известны Th-эпитопы, обладающие способностью связываться с различными молекулами MHC класса II, и можно сконструировать Th-эпитоп, который может связываться с различными молекулами MHC класса II, независимо от генетических признаков. В одном варианте осуществления Th-эпитоп может представлять собой последовательность, называемую «пан-DR-связывающий пептид», описанную в Заявке на патент США №305,871.
[00134] Конструирование Th-эпитопа 5 - исключение возможности действия в качестве B-клеточного эпитопа
[00135] Th-эпитоп предназначен для презентации молекулами MHC класса II антигенпрезентирующих клеток и распознавания хелперными Т-клетками. Таким образом, Th-эпитоп обычно обладает очень высокой способностью связывания с молекулами МНС класса II, и, таким образом, вероятность того, что Th-эпитоп может действовать как В-клеточный эпитоп, очень низка. Другими словами, Th-эпитоп разработан таким образом, что он не индуцирует антитело, которое специфически связывается с трехмерной структурой самого Th-эпитопа.
[00136] конструирование длины Th-эпитопа
[00137] Th-эпитоп должен иметь соответствующую длину, чтобы он мог связываться с одной единицей МНС класса II. Общеизвестно, что одна единица Th-эпитопа, которая может напрямую связываться с МНС класса II, имеет длину, соответствующую примерно 30-меру (Abbas, A.K., Lichtman, A.H. and Pillai, S. Cellular and molecular immunology (pp124-126), 7th Ed. (2012) Philadelphia PA, Elsevier Saunder, etc.). Кроме того, 1) когда длина Th-эпитопа становится слишком короткой, существует риск потери Th-эпитопом способности связываться с МНС класса II, тогда как 2) когда длина Th-эпитопа слишком велика, существует возможность для Th-эпитопа действовать независимо в качестве В-клеточного эпитопа, тем самым отклоняясь от заданной цели. Следовательно, необходимо сконструировать Th-эпитоп подходящей длины.
[00138] диапазон длины Th-эпитопа
[00139] В одном варианте осуществления длина Th-эпитопа может соответствовать 7-меру, 8-меру, 9-меру, 10-меру, 11-меру, 12-меру, 13-меру, 14-меру, 15-меру, 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру, 32-меру или 33-меру. В другом варианте осуществления длина Th-эпитопа может иметь значение в пределах двух числовых диапазонов, выбранных в непосредственно предшествующем предложении. Например, по длине Th-эпитоп может представлять собой от 8-мера до 32-мера. В другом примере длина Th-эпитопа может находиться в диапазоне от 11-мера до 13-мера.
[00140] варианты осуществления Th-эпитопа - конструкция PADRE
[00141] В одном варианте осуществления Th-эпитоп может представлять собой пептид, названный «пан-DR-связывающий пептид», раскрытый в заявке на патент США №305871. В другом варианте осуществления Th-эпитоп может представлять собой один из пептидов, описанных в Таблицах VIIIA и IX патента США №6413935 B1. Еще в одном варианте осуществления Th-эпитоп может иметь пептидную последовательность, удовлетворяющую следующей структурной Формуле I:
[00142][Формула I]
[00143] N-Lys-X1-X2-Ala-Ala-X3-Thr-X4-X5-Ala-Ala-C
[00144] в которой X1 может представлять собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин, но X1 не ограничивается этим.
[00145] X2 может быть гидрофобной аминокислотой или может быть лейцином (Leu) или изолейцином (Ile), но X2 этим не ограничивается.
[00146] X3 может быть ароматической или циклической аминокислотой или может быть фенилаланином (Phe), тирозином (Tyr) или гистидином (His), но X3 этим не ограничивается.
[00147] X4 может представлять собой алифатическую аминокислоту с длинной цепью или может представлять собой изолейцин (Ile) или валин (Val), но X4 не ограничивается этим.
[00148] X5 может быть заряженной аминокислотой или может быть аргинином (Arg), лейцином (Leu), аспарагиновой кислотой (Asp), глутамином (Gln) или глицином (Gly), но X5 не ограничивается этим.
[00149] В одном варианте осуществления Th-эпитоп может иметь пептидную последовательность, удовлетворяющую следующей структурной Формуле II:
[00150][Формула II]
[00151] N-X1-X2-Val-X3-Ala-X4-Thr-Leu-Lys-Ala-Ala-C
[00152] в которой X1 представляет собой лизин (Lys) или аргинин (Arg),
[00153] X2 представляет собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин,
[00154] X3 представляет собой лизин (Lys), триптофан (Trp), тирозин (Tyr), аргинин (Arg), аланин (Ala) или метионин (Met), и
[00155] X4 представляет собой аспарагин (Asn), триптофан (Trp), тирозин (Tyr), валин (Val), гистидин (His), лизин (Lys) или аланин (Ala).
[00156] варианты последовательностей Th-эпитопа
[00157] В одном варианте осуществления Th-эпитоп может быть выбран из группы, состоящей из K(Cha)VAAWTLKAA(SEQ ID NO: 1), PKYVKQNTLKLAT(SEQ ID NO: 2), ILMQYIKANSKFIGI(SEQ ID NO: 3), QSIALSSLMVAQAIP(SEQ ID NO: 4), ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ(SEQ ID NO: 5), PLGFFPDHQL(SEQ ID NO: 162), WPEANQVGAGAFGPGF(SEQ ID NO: 163), MQWNSTALHQALQDP(SEQ ID NO: 164), MQWNSTTFHQTLQDPRVRGLYFPAGG(SEQ ID NO: 165), FFLLTRILTI(SEQ ID NO: 166), FFLLTRILTIPQSLD(SEQ ID NO: 167), TSLNFLGGTTVCLGQ(SEQ ID NO: 168), 17 QSPTSNHSPTSCPPIC(SEQ ID NO: 169), IIFLFILLLCLIFLLVLLD(SEQ ID NO: 170), CTTPAQGNSMFPSC(SEQ ID NO: 171), CTKPTDGN(SEQ ID NO: 172), WASVRFSW(SEQ ID NO: 173), LLPIFFCLW(SEQ ID NO: 174), MDIDPYKEFGATVELLSFLP(SEQ ID NO: 175), FLPSDFFPSV(SEQ ID NO: 176), RDLLDTASALYREALESPEH(SEQ ID NO: 177), PHHTALRQAILCWGELMTLA(SEQ ID NO: 178), GRETVIEYLVSFGVW(SEQ ID NO: 179), EYLVSFGVWIRTPPA(SEQ ID NO: 180), VSFGVWIRTPPAYRPPNAPI(SEQ ID NO: 181), TVVRRRGRSP(SEQ ID NO: 182), VGPLTVNEKRRLKLI(SEQ ID NO: 183), RHYLHTLWKAGILYK(SEQ ID NO: 184), ESRLVVDFSQFSRGN(SEQ ID NO: 185), LQSLTNLLSSNLSWL(SEQ ID NO: 186), SSNLSWLSLDVSAAF(SEQ ID NO: 187), LHLYSHPIILGFRKI(SEQ ID NO: 188), KQCFRKLPVNRPIDW(SEQ ID NO: 189), LCQVFADATPTGWGL(SEQ ID NO: 190), AANWILRGTSFVYVP(SEQ ID NO: 191), EIRLKVFVLGGCRHK(SEQ ID NO: 192), KFVAAWTLKAA(SEQ ID NO: 195), KYVAAWTLKAA(SEQ ID NO: 196), DIEKKIAKMEKASSVFNVVNS(SEQ ID NO: 223), YSGPLKAEIAQRLEDV(SEQ ID NO: 224), K(Cha)VKANTLKAA(SEQ ID NO: 225), K(Cha)VKANTLKAA(SEQ ID NO: 226), K(Cha)VKAWTLKAA(SEQ ID NO: 227), K(Cha)VKAWTLKAA(SEQ ID NO: 228), K(Cha)VWANTLKAA(SEQ ID NO: 229), K(Cha)VWANTLKAA(SEQ ID NO: 230), K(Cha)VWAYTLKAA(SEQ ID NO: 231), K(Cha)VWAVTLKAA(SEQ ID NO: 232), K(Cha)VYAWTLKAA(SEQ ID NO: 233), K(Cha)VYAWTLKAA(SEQ ID NO: 234), R(Cha)VRANTLKAA(SEQ ID NO: 235), K(Cha)VKAHTLKAA(SEQ ID NO: 236), K(Cha)VKAHTLKAA(SEQ ID NO: 237), K(Cha)VAANTLKAA(SEQ ID NO: 238), K(Cha)VAANTLKAA(SEQ ID NO: 239), K(Cha)VAAYTLKAA(SEQ ID NO: 240), K(Cha)VAAYTLKAA(SEQ ID NO: 241), K(Cha)VAAWTLKAA(SEQ ID NO: 242), K(Cha)VAAKTLKAA(SEQ ID NO: 243), K(Cha)VAAHTLKAA(SEQ ID NO: 244), K(Cha)VAAATLKAA(SEQ ID NO: 245), K(Cha)VAAWTLKAA(SEQ ID NO: 246), и K(Cha)VMAATLKAA(SEQ ID NO: 247). В этом случае «а» обозначает D-форму аланина, «Z» обозначает 6-аминогексановую кислоту, а «(Cha)» обозначает L-циклогексилаланин.
[00158] последовательности, сходные с типичными последовательностями Th-эпитопа
[00159] В настоящем описании раскрыты последовательности, сходные с типичными последовательностями Th-эпитопа. В одном варианте осуществления Th-эпитоп может иметь последовательность, имеющую 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с SEQ ID NO: 1 - SEQ ID NO: 5, SEQ ID NO: 162 - SEQ ID NO: 192, SEQ ID NO: 195 - SEQ ID NO: 196 и SEQ ID NO: 223 - SEQ ID NO: 247, последовательность, удовлетворяющую приведенной выше[Формуле I], или последовательность, удовлетворяющую приведенной выше[Формуле II]. В другом варианте осуществления Th-эпитоп может иметь последовательность, которая соответствует по номеру, выбранному в непосредственно предшествующем предложении или более, SEQ ID NO: 1 - SEQ ID NO: 5, SEQ ID NO: 162 - SEQ ID NO: 192, SEQ ID NO: 195 - SEQ ID NO: 196 и SEQ ID NO: 223 - SEQ ID NO: 247, последовательность, удовлетворяющую приведенной выше[Формуле I], или последовательность, удовлетворяющую приведенной выше[Формуле II]. Например, Th-эпитоп может иметь последовательность, которая на 90% или более идентична последовательности SEQ ID NO: 1.
[00160] ВСПОМОГАТЕЛЬНАЯ ЧАСТЬ
[00161] определение вспомогательной части
[00162] Описанные в настоящем документе пептиды могут включать одну или более вспомогательных частей. Вспомогательная часть в совокупности относится к дополнительной части, которая может прямо или косвенно воздействовать на пептид, вызывая заданный иммунный ответ в организме пациента. Вспомогательная часть может иметь одну или более функций, и состав пептидной последовательности и/или положение последовательности могут быть соответствующим образом сконструированы в соответствии с целью.
[00163] функция вспомогательной части 1 - функция в качестве линкера
[00164] Вспомогательная часть может функционировать в качестве линкера, связывающего В-клеточный эпитоп и Th-эпитоп.В-клеточный эпитоп и Th-эпитоп могут быть связаны напрямую или они могут быть связаны через вспомогательную часть, которая служит линкером. Кроме того, вспомогательная часть может иметь линкерную функцию, которая связывает множество единиц, включенных в пептид. В одном варианте осуществления последовательность вспомогательной части может быть расположена между последовательностью В-клеточного эпитопа и последовательностью Th-эпитопа. В частности, вспомогательная часть выполняет линкерную функцию, который связывает В-клеточный эпитоп и Th-эпитоп.В другом варианте осуществления последовательность вспомогательной части может быть расположена между последовательностью первой пептидной единицы и последовательностью второй пептидной единицы в пептиде. В частности, вспомогательная часть выполняет линкерную функцию для связывания первой пептидной единицы и второй пептидной единицы.
[00165] функция вспомогательной части 2 - функция защиты
[00166] В случае пептидной единицы, предложенной в настоящем описании, она характеризуется относительно короткой длиной последовательности. Соответственно, когда пептид, включающий пептидную единицу, вводят инъекцией в организм пациента, последовательность Th-эпитопа может быть разрушена до того, как будет распознана хелперными Т-клетками, и, таким образом, предполагаемый иммунный ответ может не возникнуть. В одном варианте осуществления защитная единица может защищать Th-эпитоп от расщепления ферментом в организме пациента. Например, фермент в организме пациента может представлять собой пептидазу. В частности, пептидаза может представлять собой экзопептидазу и/или эндопептидазу, но пептидаза этим не ограничивается. В другом варианте осуществления вспомогательная часть может быть связана с N-концом и/или С-концом Th-эпитопа. В частности, вспомогательная часть выполняет функцию защиты Th-эпитопа. Еще в одном варианте вспомогательная часть может включать по меньшей мере одну нестандартную аминокислоту.
[00167] функция вспомогательной части 3 - функция образования циклической формы
[00168] Вспомогательная часть может быть сконструирована таким образом, чтобы соединяться с обоими концами пептидной единицы, чтобы таким образом выполнять функцию, позволяющую пептиду образовывать циклическую форму. В одном варианте осуществления пептид может включать первую вспомогательную часть на N-конце и вторую вспомогательную часть на С-конце. В частности, каждая из первой вспомогательной части и второй вспомогательной части может включать один или более цистеинов (S). В другом варианте осуществления пептид может существовать в циклической форме. В частности, N-конец и С-конец пептида могут быть связаны через вспомогательную часть.
[00169] функция вспомогательной части 3 - другие функции
[00170] Вспомогательная часть может иметь дополнительную функцию в дополнение к вышеперечисленным функциям. В одном варианте осуществления вспомогательная часть может включать гидрофильную аминокислоту и может выполнять функцию повышения растворимости пептида. В другом варианте осуществления вспомогательная часть может состоять из последовательности, биологически неактивной в организме пациента. В частности, вспомогательная часть не влияет на функции В-клеточного эпитопа и Th-эпитопа и может иметь фиктивную функцию для увеличения длины пептида. В частности, пептид может представлять собой His-метку, но не ограничивается этим.
[00171] способность выполнять несколько функций
[00172] Вспомогательная часть может иметь одну или более функций. В одном варианте осуществления вспомогательная часть может иметь линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости.
[00173] может включать нестандартную аминокислоту
[00174] Вспомогательная часть может включать одну или более нестандартных аминокислот.Искусственная аминокислота может быть необходима для того, чтобы вспомогательная часть проявляла линкерную функцию, защитную функцию и/или другие функции. В одном варианте осуществления вспомогательная часть может включать по меньшей мере одну нестандартную аминокислоту. В частности, нестандартная аминокислота может представлять собой одну или более нестандартных аминокислот, выбранных из группы, состоящей из L-циклогексилаланина, D-формы аланина и 6-аминогексановой кислоты, но нестандартная аминокислота этим не ограничивается.
[00175] длина вспомогательной части
[00176] Вспомогательная часть может иметь соответствующую длину в зависимости от ее функции. Когда вспомогательная часть выполняет несколько функций, она может иметь соответствующую длину для выполнения всех этих функций. В одном варианте осуществления длина вспомогательной части может соответствовать 1-меру, 2-меру, 3-меру, 4-меру, 5-меру, 6-меру, 7-меру, 8-меру, 9-меру, 10-меру, 11-меру, 12-меру, 13-меру, 14-меру, 15-меру, 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру или 31-меру или длиннее. В другом варианте осуществления длина вспомогательной части может иметь значение в пределах двух числовых диапазонов непосредственно предшествующего предложения. Например, длина вспомогательной части может быть в диапазоне от 1-мера до 8-мера. В другом примере длина вспомогательной части может быть в диапазоне от 15-мера до 26-мера.
[00177] характеристики вспомогательной части - мало влияет на функцию В-клеточного эпитопа
[00178] Вспомогательная часть не оказывает существенного влияния на функцию пептидной единицы и/или пептида, раскрытых в настоящем описании, индуцировать антитело, которое специфически связывается с В-клеточным эпитопом в организме пациента.
[00179] варианты последовательностей вспомогательной части
[00180] В одном варианте осуществления вспомогательная часть может представлять собой пептид, выбранный из группы, состоящей из a, Z, aZ, Za, RN, AF, CR, LS, KT, KH, RF, DP, SV, GL, ZRNV(SEQ ID NO: 36), aZRN(SEQ ID NO: 37), IAFZ(SEQ ID NO: 38), AFZa(SEQ ID NO: 39), RNVP(SEQ ID NO: 40), WIAF(SEQ ID NO: 41), ZCRF(SEQ ID NO: 42), aZCR(SEQ ID NO: 43), YLSZ(SEQ ID NO: 44), LSZa(SEQ ID NO: 45), CRFR(SEQ ID NO: 46), VYLS(SEQ ID NO: 47), ZKTT(SEQ ID NO: 48), aZKT(SEQ ID NO: 49), NKHZ(SEQ ID NO: 50), KHZa(SEQ ID NO: 51), GSHHHHHHGSDDDDK(SEQ ID NO: 52), HHHHHH(SEQ ID NO: 53), MRGSHHHHHHGSDDDDKIVD(SEQ ID NO: 54), GGGGSGGGGGGSS(SEQ ID NO: 55), RRRRRR(SEQ ID NO: 159), GSHHHHHHGSDDDDKaZ(SEQ ID NO: 193), и ZaGSHHHHHHGSDDDDK(SEQ ID NO: 194). В частности, «а» обозначает D-форму аланина, а «Z» обозначает 6-аминогексановую кислоту.
[00181] КОНСТРУИРОВАНИЕ ПЕПТИДНОЙ ЕДИНИЦЫ - ОБЩЕЕ
[00182] Способ конструирования возможной пептидной единицы и ее формы будут описаны ниже. Каждая единица может включать по меньшей мере один В-клеточный эпитоп и по меньшей мере один Th-эпитоп и может включать соответствующее количество вспомогательных частей. Порядок связывания В-клеточного эпитопа, Th-эпитопа и вспомогательной части приведен в качестве примера для каждого типа. Если не указано иное, конструкция каждой части, включенной в пептидную единицу, в основном соответствует принципу конструкции, описанному выше.
[00183] КОНСТРУКЦИЯ ЕДИНИЦЫ-А
[00184] структура единицы-А 1 - обзор
[00185] В качестве пептидной единицы, предложенной в настоящем описании, пептидная единица, которая может включать 1) один В-клеточный эпитоп и один Th-эпитоп и 2) одну или более вспомогательных частей, называется «единицей-А». Функция вспомогательной части конкретно не ограничена, пока она не нарушает функции В-клеточного эпитопа и Th-эпитопа, и при необходимости сконструирована надлежащим образом.
[00186] В одном варианте осуществления единица-А может представлять собой единицу, в которой первый В-клеточный эпитоп и первый Th-эпитоп последовательно связаны в направлении от N-конца к С-концу.
[00187] Кроме того, единица-А может дополнительно включать первую вспомогательную часть. Когда единица-А включает первую вспомогательную часть, последовательность первой вспомогательной части расположена в N-концевой области относительно последовательности первого В-клеточного эпитопа в последовательности единицы-А. В частности, первая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции первой вспомогательной части не ограничиваются этим.
[00188] Кроме того, единица-А может дополнительно включать вторую вспомогательную часть. Когда единица-А включает вторую вспомогательную часть, последовательность второй вспомогательной части расположена между последовательностью первого В-клеточного эпитопа и последовательностью первого Th-эпитопа внутри последовательности единицы-А. В частности, вторая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции второй вспомогательной части не ограничиваются этим.
[00189] Кроме того, единица-А может дополнительно включать третью вспомогательную часть. Когда единица-А включает третью вспомогательную часть, последовательность третьей вспомогательной части расположена в С-концевой области относительно последовательности первого Th-эпитопа в последовательности единицы-А. В частности, третья вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию, защитную функцию и/или функцию образования циклической формы, но функции третьей вспомогательной части не ограничиваются этим.
[00190] В другом варианте осуществления единица-А может представлять собой единицу, в которой второй Th-эпитоп и второй В-клеточный эпитоп последовательно связаны в направлении от N-конца к С-концу.
[00191] Кроме того, единица-А может дополнительно включать четвертую вспомогательную часть. Когда единица-А включает четвертую вспомогательную часть, последовательность четвертой вспомогательной части расположена в N-концевой области относительно последовательности второго Th-эпитопа в последовательности единицы-А. В частности, четвертая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции четвертой вспомогательной части не ограничиваются этим.
[00192] Кроме того, единица-А может дополнительно включать пятую вспомогательную часть. Когда единица-А включает пятую вспомогательную часть, последовательность пятой вспомогательной части расположена между последовательностью второго В-клеточного эпитопа и последовательностью второго Th-эпитопа внутри последовательности единицы-А. В частности, пятая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции пятой вспомогательной части не ограничиваются этим.
[00193] Кроме того, единица-А может дополнительно включать шестую вспомогательную часть. Когда единица-А включает шестую вспомогательную часть, последовательность шестой вспомогательной части расположена в C-концевой области относительно последовательности второго В-клеточного эпитопа в последовательности единицы-А. В частности, шестая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции шестой вспомогательной части не ограничиваются этим.
[00194] структура единицы-А 2 - формула
[00195] В одном варианте осуществления единица-А представляет собой пептид, представленный следующей [Формулой А] или [Формулой А'].
[00196] [Формула А]
[00197] N-А1-В1-А2-Т1-А3-С
[00198] [Формула А']
[00199] N-А4-Т2-А5-В2-А6-С
[00200] B1 и B2 представляют собой B-клеточные эпитопы, и они следуют принципу конструкции, описанному выше.
[00201] T1 и T2 представляют собой Th-эпитопы, и они следуют принципу конструкции, описанному выше.
[00202] A1-A6 представляют собой вспомогательные части и могут быть опущены.
[00203] В частности, A1-A6 могут иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но A1-A6 этим не ограничиваются.
[00204] длина единицы-А
[00205] В одном варианте осуществления длина единицы-А может соответствовать примерно 16-меру, примерно 17-меру, примерно 18-меру, примерно 19-меру, примерно 20-меру, примерно 21-меру, примерно 22-меру, примерно 23-меру, примерно 24-меру, примерно 25-меру, примерно 26-меру, примерно 27-меру, примерно 28-меру, примерно 29-меру, примерно 30-меру, примерно 31-меру, примерно 32-меру, примерно 33-меру, примерно 34-меру, примерно 35-меру, примерно 36-меру, примерно 37-меру, примерно 38-меру, примерно 39-меру, примерно 40-меру, примерно 41-меру, примерно 42-меру, примерно 43-меру, примерно 44-меру, примерно 45-меру, примерно 46-меру, примерно 47-меру, примерно 48-меру, примерно 49-меру, примерно 50-меру, примерно 51-меру, примерно 52-меру, примерно 53-меру, примерно 54-меру, примерно 55-меру, примерно 56-меру, примерно 57-меру, примерно 58-меру, примерно 59-меру, примерно 60-меру, примерно 61-меру, примерно 62-меру, примерно 63-меру, примерно 64-меру, примерно 65-меру, примерно 66-меру, примерно 67-меру, примерно 68-меру, примерно 69-меру, примерно 70-меру, примерно 71-меру, примерно 72-меру, примерно 73-меру, примерно 74-меру, примерно 75-меру, примерно 76-меру, примерно 77-меру, примерно 78-меру, примерно 79-меру, примерно 80-меру, примерно 81-меру, примерно 82-меру, примерно 83-меру, примерно 84-меру, примерно 85-меру, примерно 86-меру, примерно 87-меру, примерно 88-меру, примерно 89-меру, примерно 90-меру, примерно 91-меру, примерно 92-меру, примерно 93-меру, примерно 94-меру, примерно 95-меру, примерно 96-меру, примерно 97-меру, примерно 98-меру, примерно 99-меру или примерно 100-меру. В другом варианте осуществления длина единицы-А может иметь значение в пределах двух числовых диапазонов, выбранных в непосредственно предшествующем предложении. Например, длина единицы-А может находиться в диапазоне от примерно 16-мера до примерно 30-мера. В другом примере длина единицы-А может находиться в диапазоне от примерно 23-мера до примерно 60-мера.
[00206] вариант единицы А - примерная конструкция
[00207] В одном варианте осуществления единица-А может иметь последовательность, в которой последовательно связаны первый В-клеточный эпитоп, первая вспомогательная часть и первый Th-эпитоп.В частности, первая вспомогательная часть выполняет линкерную функцию и включает одну или более искусственных аминокислот.
[00208] В другом варианте осуществления единица-А может иметь последовательность, в которой последовательно связаны вторая вспомогательная часть, второй В-клеточный эпитоп, третья вспомогательная часть и второй Th-эпитоп.В частности, вторая вспомогательная часть представляет собой His-метку, а третья вспомогательная часть выполняет линкерную функцию и включает одну или более искусственных аминокислот.
[00209] Еще в одном варианте осуществления единица-А может иметь последовательность, в которой последовательно связаны третий В-клеточный эпитоп, четвертая вспомогательная часть, третий Th-эпитоп и пятая вспомогательная часть. В частности, четвертая вспомогательная часть выполняет линкерную функцию и защитную функцию, а пятая вспомогательная часть выполняет защитную функцию. Каждая из четвертой вспомогательной части и пятой вспомогательной части включает одну или более искусственных аминокислот.
[00210] Еще в одном варианте осуществления единица-А может иметь последовательность, в которой последовательно связаны 6-я вспомогательная часть, 4-й В-клеточный эпитоп, 7-я вспомогательная часть, 4-й Th-эпитоп и 8-я вспомогательная часть. В частности, шестая вспомогательная часть представляет собой His-метку, седьмая вспомогательная часть выполняет линкерную функцию и защитную функцию, а восьмая вспомогательная часть выполняет защитную функцию. Седьмая вспомогательная часть и восьмая вспомогательная часть включают одну или более искусственных аминокислот.
[00211] варианты осуществления последовательности единицы-А
[00212] В одном варианте осуществления единица-A представляет собой пептидную единицу, выбранную из группы, состоящей из RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 56), ZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF(SEQ ID NO: 57), CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 58), ZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLS(SEQ ID NO: 59), KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 60), ZaK(Cha)VAAWTLKAAaZKTTKQSFDLSVKAQYKKNKH(SEQ ID NO: 61), RNVPPIFNDVYWIAFK(Cha)VAAWTLKAA(SEQ ID NO: 62), K(Cha)VAAWTLKAARNVPPIFNDVYWIAF(SEQ ID NO: 63), RNVPPIFNDVYK(Cha)VAAWTLKAA(SEQ ID NO: 64), PIFNDVYWIAFK(Cha)VAAWTLKAA(SEQ ID NO: 65), PPIFNDVYWK(Cha)VAAWTLKAA(SEQ ID NO: 66), RNVPPIFNDVYWIAFK(Cha)VAAWTLKAAHHHHHH(SEQ ID NO: 67), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZGSHHHHHHGSDDDDK(SEQ ID NO: 68), GSHHHHHHGSDDDDKZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF(SEQ ID NO: 69), RNVPPIFNDVYWIAFGSHHHHHHGSDDDDKZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 70), GSHHHHHHGSDDDDKZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLS(SEQ ID NO: 71), GSHHHHHHGSDDDDKCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 72), CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZGSHHHHHHGSDDDDK(SEQ ID NO: 73), GSHHHHHHGSDDDDKZaK(Cha)VAAWTLKAAaZKTTKQSFDLSVKAQYKKNK H(SEQ ID NO: 74), GSHHHHHHGSDDDDKKTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAA aZ(SEQ ID NO: 75), KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZGSHHHHHHGSDDDD K(SEQ ID NO: 76), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 77), MRGSHHHHHHGSDDDDKIVDGSHHHHHHGSDDDDKRNVPPIFNDVYWIAFZa K(Cha)VAAWTLKAAaZ(SEQ ID NO: 78), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ GSHHHHHHGSDDDDK(SEQ ID NO: 79), RNVPPIFNDVYWIAFILMQYIKANSKFIGI(SEQ ID NO: 80), RNVPPIFNDVYWIAFILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ(SEQ ID NO: 81), CRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZC(SEQ ID NO: 82), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAACR(SEQ ID NO: 161), RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXGSHHHHHHGSDDDDK(SEQ ID NO: 199), GSHHHHHHGSDDDDKXXKXVAAWTLKAAXXRNVPPIFNDVYWIAF(SEQ ID NO: 200), RNVPPIFNDVYWIAFXXKXVAAWTLKAAXX(SEQ ID NO: 204), RNVPPIFNDVYWIAFKXVAAWTLKAA(SEQ ID NO: 205), RNVPPIFNDVYWIAFKXVAAWTLKAAHHHHHH(SEQ ID NO: 206), RNVPPIFNDVYWIAFXXKXVAAWTLKAACR(SEQ ID NO: 208), RNVPPIFNDVYWIAFXXKFVAAWTLKAAXX(SEQ ID NO: 210), RNVPPIFNDVYWIAFXXKFVAAWTLKAACR(SEQ ID NO: 212), RNVPPIFNDVYWIAFCTKPTDGN(SEQ ID NO: 213), RNVPPIFNDVYWIAFLLPIFFCLW(SEQ ID NO: 214), RNVPPIFNDVYWIAFFLPSDFFPSV(SEQ ID NO: 215), RNVPPIFNDVYWIAFILMQYIKANSKFIGIHHHHHH(SEQ ID NO: 219), и RNVPPIFNDVYWIAFMDIDPYKEFGATVELLSFLPHHHHHH(SEQ ID NO: 220). В этом случае «а» обозначает D-форму аланина, «Z» обозначает 6-аминогексановую кислоту, «(Cha)» обозначает L-циклогексилаланин, и «X» обозначает любую стандартную аминокислоту.
[00213] КОНСТРУКЦИЯ ЕДИНИЦЫ-B
[00214] структура единицы-B 1 - обзор
[00215] В качестве пептидной единицы, предложенной в настоящем документе, пептидная единица 1), которая включает два В-клеточных эпитопа и один Th-эпитоп, 2) в которой последовательность одного В-клеточного эпитопа из двух В-клеточных эпитопов расположена между последовательностью другого В-клеточного эпитопа и последовательностью Th-эпитопа, и 3), которая может включать одну или более вспомогательных частей, называется «единицей-В». Функция вспомогательной части конкретно не ограничена, пока она не нарушает функции В-клеточного эпитопа и Th-эпитопа, и при необходимости сконструирована надлежащим образом.
[00216] В одном варианте осуществления единица-В может представлять собой единицу, в которой первый В-клеточный эпитоп, второй В-клеточный эпитоп и первый Th-эпитоп последовательно связаны в направлении от N-конца к С-концу.
[00217] Кроме того, единица-В может дополнительно включать первую вспомогательную часть. Когда единица-В включает первую вспомогательную часть, последовательность первой вспомогательной части расположена в N-концевой области относительно последовательности первого В-клеточного эпитопа в последовательности единицы-В. В частности, первая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции первой вспомогательной части не ограничиваются этим.
[00218] Кроме того, единица-В может дополнительно включать вторую вспомогательную часть. Когда единица-В включает вторую вспомогательную часть, последовательность второй вспомогательной части расположена между последовательностью первого В-клеточного эпитопа и последовательностью второго В-клеточного эпитопа в последовательности единицы-В. В частности, вторая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции второй вспомогательной части не ограничиваются этим.
[00219] Кроме того, единица-В может дополнительно включать третью вспомогательную часть. Когда единица-В включает третью вспомогательную часть, последовательность третьей вспомогательной части расположена между последовательностью второго В-клеточного эпитопа и последовательностью первого Th-эпитопа в последовательности единицы-В. В частности, третья вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции третьей вспомогательной части не ограничиваются этим.
[00220] Кроме того, единица-В может дополнительно включать четвертую вспомогательную часть. Когда единица-В включает четвертую вспомогательную часть, последовательность четвертой вспомогательной части расположена в C-концевой области относительно последовательности первого Th-эпитопа в последовательности единицы-В. В частности, четвертая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции четвертой вспомогательной части не ограничиваются этим.
[00221] В другом варианте осуществления единица-В может представлять собой единицу, в которой второй Th-эпитоп, третий В-клеточный эпитоп и четвертый В-клеточный эпитоп последовательно связаны в направлении от N-конца к С-концу.
[00222] Кроме того, единица-В может дополнительно включать пятую вспомогательную часть. Когда единица-В включает пятую вспомогательную часть, последовательность пятой вспомогательной части расположена в N-концевой области относительно последовательности второго Th-эпитопа в последовательности единицы-В. В частности, пятая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции пятой вспомогательной части не ограничиваются этим.
[00223] Кроме того, единица-В может дополнительно включать шестую вспомогательную часть. Когда единица-В включает шестую вспомогательную часть, последовательность шестой вспомогательной части расположена между последовательностью второго Th-эпитопа и последовательностью третьего В-клеточного эпитопа внутри последовательности единицы-В. В частности, шестая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции шестой вспомогательной части не ограничиваются этим.
[00224] Кроме того, единица-В может дополнительно включать седьмую вспомогательную часть. Когда единица-В включает седьмую вспомогательную часть, последовательность седьмой вспомогательной части располагается между последовательностью третьего В-клеточного эпитопа и последовательностью четвертого В-клеточного эпитопа в последовательности единицы-В. В частности, седьмая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции седьмой вспомогательной части не ограничиваются этим.
[00225] Кроме того, единица-В может дополнительно включать восьмую вспомогательную часть. Когда единица-В включает восьмую вспомогательную часть, последовательность восьмой вспомогательной части расположена в C-концевой области относительно последовательности четвертого В-клеточного эпитопа в последовательности единицы-В. В частности, восьмая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции восьмой вспомогательной части не ограничиваются этим.
[00226] структура единицы-B 2 - формула
[00227] В одном варианте осуществления единица-В представляет собой пептид, представленный следующей [Формулой В] или [Формулой В'].
[00228] [Формула B]
[00229] N-А1-В1-А2-В2-А3-Т1-А4-С
[00230] [Формула B']
[00231] N-A5-T2-A6-B3-A7-B4-A8-C
[00232] B1-B4 представляют собой B-клеточные эпитопы, и они следуют принципу конструкции, описанному выше.
[00233] T1 и T2 представляют собой Th-эпитопы, и они следуют принципу конструкции, описанному выше.
[00234] A1-A8 представляют собой вспомогательные части и могут быть опущены.
[00235] В частности, A1-A8 могут иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но A1-A8 этим не ограничиваются.
[00236] длина единицы-B
[00237] В одном варианте осуществления длина единицы-В может соответствовать примерно 24-меру, примерно 25-меру, примерно 26-меру, примерно 27-меруа, примерно 28-меру, примерно 29-меру, примерно 30-меру, примерно 31-меру, примерно 32-меру, примерно 33-меру, примерно 34-меру, примерно 35-меру, примерно 36-меру, примерно 37-меру, примерно 38-меру, примерно 39-меру, примерно 40-меру, примерно 41-меру, примерно 42-меру, примерно 43-меру, примерно 44-меру, примерно 45-меру, примерно 46-меру, примерно 47-меру, примерно 48-меру, примерно 49-меру, примерно 50-меру, примерно 51-меру, примерно 52-меру, примерно 53-меру, примерно 54-меру, примерно 55-меру, примерно 56-меру, примерно 57-меру, примерно 58-меру, примерно 59-меру, примерно 60-меру, примерно 61-меру, примерно 62-меру, примерно 63-меру, примерно 64-меру, примерно 65-меру, примерно 66-меру, примерно 67-меру, примерно 68-меру, примерно 69-меру, примерно 70-меру, примерно 71-меру, примерно 72-меру, примерно 73-меру, примерно 74-меру, примерно 75-меру, примерно 76-меру, примерно 77-меру, примерно 78-меру, примерно 79-меру, примерно 80-меру, примерно 81-меру, примерно 82-меру, примерно 83-меру, примерно 84-меру, примерно 85-меру, примерно 86-меру, примерно 87-меру, примерно 88-меру, примерно 89-меру, примерно 90-меру, примерно 91-меру, примерно 92-меру, примерно 93-меру, примерно 94-меру, примерно 95-меру, примерно 96-меру, примерно 97-меру, примерно 98-меру, примерно 99-меру или примерно 100-меру. В другом варианте осуществления длина единицы-В может иметь значение в пределах двух числовых диапазонов, выбранных в непосредственно предшествующем предложении. Например, длина единицы-В может находиться в диапазоне от примерно 24-мера до примерно 45-мера. В другом примере длина единицы-А может находиться в диапазоне от примерно 40-мера до примерно 80-мера.
[00238] вариант осуществления единицы-B - примерная конструкция
[00239] В одном варианте осуществления единица-В может иметь последовательность, в которой последовательно связаны первая вспомогательная часть, первый В-клеточный эпитоп, второй В-клеточный эпитоп, вторая вспомогательная часть и первый Th-эпитоп.В частности, первая вспомогательная часть представляет собой His-метку, вторая вспомогательная часть включает одну или более искусственных аминокислот.
[00240] В другом варианте осуществления единица-В может иметь последовательность, в которой последовательно связаны третий В-клеточный эпитоп, четвертый В-клеточный эпитоп, третья вспомогательная часть и второй Th-эпитоп.В частности, третья вспомогательная часть выполняет линкерную функцию и включает одну или более искусственных аминокислот.
[00241] В другом варианте осуществления единица-В может иметь последовательность, в которой четвертая вспомогательная часть, пятый В-клеточный эпитоп, шестой В-клеточный эпитоп, пятая вспомогательная часть, третий Th-эпитоп и шестая вспомогательная часть последовательно связаны. В частности, четвертая вспомогательная часть представляет собой His-метку, пятая вспомогательная часть выполняет линкерную функцию и защитную функцию и включает одну или более искусственных аминокислот, а шестая вспомогательная часть выполняет защитную функцию и включает одну или более искусственных аминокислот.
[00242] Еще в одном варианте осуществления единица-В может иметь последовательность, в которой последовательно связаны седьмой В-клеточный эпитоп, восьмой В-клеточный эпитоп, седьмая вспомогательная часть, четвертый Th-эпитоп и восьмая вспомогательная часть. В частности, седьмая вспомогательная часть имеет линкерную функцию, защитную функцию и включает одну или более искусственных аминокислот, а восьмая вспомогательная часть выполняет защитную функцию и включает одну или более искусственных аминокислот.
[00243] Еще в одном варианте осуществления единица-В может иметь последовательность, в которой восьмая вспомогательная часть, девятый В-клеточный эпитоп, девятая вспомогательная часть, десятый В-клеточный эпитоп, десятая вспомогательная часть и пятый Th-эпитоп связаны последовательно. В частности, восьмая вспомогательная часть представляет собой His-метку, девятая вспомогательная часть выполняет линкерную функцию, а десятая вспомогательная часть выполняет линкерную функцию и включает одну или более искусственных аминокислот.
[00244] Еще в одном варианте осуществления единица-В может иметь последовательность, в которой последовательно связаны одиннадцатый В-клеточный эпитоп, одиннадцатая вспомогательная часть, двенадцатый В-клеточный эпитоп, двенадцатая вспомогательная часть и шестой Th-эпитоп.В частности, одиннадцатая вспомогательная часть выполняет линкерную функцию, а двенадцатая вспомогательная часть выполняет линкерную функцию и включает одну или более искусственных аминокислот.
[00245] Еще в одном варианте осуществления единица-В может иметь последовательность, в которой тринадцатый В-клеточный эпитоп, тринадцатая вспомогательная часть, четырнадцатый В-клеточный эпитоп, четырнадцатая вспомогательная часть, 7-й Th-эпитоп и пятнадцатая вспомогательная часть последовательно связаны. В частности, тринадцатая вспомогательная часть выполняет линкерную функцию, четырнадцатая вспомогательная часть выполняет линкерную функцию и защитную функцию и включает одну или более искусственных аминокислот, а пятнадцатая вспомогательная часть выполняет защитную функцию и включает одну или более искусственных аминокислот.
[00246] варианты осуществления последовательности единицы-B
[00247] В одном варианте осуществления единица-В представляет собой пептидную единицу, выбранную из группы, состоящей из RNVPPIFNDVYWIAFRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 83), RNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 84), RNVPPIFNDVYWIAFKTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 85), CRFRGLISLSQVYLSRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 86), CRFRGLISLSQVYLSCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 87), CRFRGLISLSQVYLSKTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 88), KTTKQSFDLSVKAQYKKNKHRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 89), KTTKQSFDLSVKAQYKKNKHCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 90), KTTKQSFDLSVKAQYKKNKHKTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWT LKAAaZ(SEQ ID NO: 91), RNVPPIFNDVYWIAFCRFRGLISLSQVYLSK(Cha)VAAWTLKAA(SEQ ID NO: 92), PIFNDVYWIAFGLISLSQVYLSK(Cha)VAAWTLKAA(SEQ ID NO: 93), RNVPPIFNDVYCRFRGLISLSQK(Cha)VAAWTLKAA(SEQ ID NO: 94), PIFNDVYWIAFCRFRGLISLSQK(Cha)VAAWTLKAA(SEQ ID NO: 95), PPIFNDVYWRGLISLSQVK(Cha)VAAWTLKAA(SEQ ID NO: 96), RNVPPIFNDVYWIAFCRFRGLISLSQVYLSK(Cha)VAAWTLKAAHHHHHH(SEQ ID NO: 97), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 98), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAA(SEQ ID NO: 99), RNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAA(SEQ ID NO: 100), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSRNVP PIFNDVYWIAFZaK(Cha)VAAWTLKAA(SEQ ID NO: 101), RNVPPIFNDVYWIAFGGGGSGGGGGGSSRNVPPIFNDVYWIAFZaK(Cha)VAA WTLKAA(SEQ ID NO: 102), RNVPPIFNDVYWIAFGGGGSGGGGGGSSRNVPPIFNDVYWIAFZaK(Cha)VAA WTLKAAaZ(SEQ ID NO: 103), RNVPPIFNDVYWIAFRNVPPIFNDVYWIAFILMQYIKANSKFIGI(SEQ ID NO: 104), RNVPPIFNDVYWIAFRNVPPIFNDVYWIAFILMQYIKANSKFIGIPMGLPQSIALS SLMVAQ(SEQ ID NO: 105), CRNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZC(SEQ ID NO: 106), и RNVPPIFNDVYWIAFCRFRGLISLSQVYLSXXK(Cha)VAAWTLKAAXX(SEQ ID NO: 202). В этом случае «а» обозначает D-форму аланина, «Z» обозначает 6-аминогексановую кислоту, «(Cha)» обозначает L-циклогексилаланин, и «X» обозначает любую стандартную аминокислоту.
[00248] КОНСТРУКЦИЯ ЕДИНИЦЫ-C
[00249] структура единицы-C 1 - обзор
[00250] В качестве пептидной единицы, предложенной в настоящем описании, пептидная единица 1), включающая два В-клеточных эпитопа и один Th-эпитоп, 2) в которой последовательность Th-эпитопа расположена между последовательностями одного В-клеточного эпитопа из двух В-клеточных эпитопов и последовательностью другого В-клеточного эпитопа, и 3) которая может включать одну или более вспомогательных частей, называется «единица-С». Функция вспомогательной части конкретно не ограничена, пока она не нарушает функции В-клеточного эпитопа и Th-эпитопа, и при необходимости сконструирована надлежащим образом.
[00251] В одном варианте осуществления единица-С может представлять собой единицу, в которой первый В-клеточный эпитоп, первый Th-эпитоп и второй В-клеточный эпитоп последовательно связаны в направлении от N-конца к С-концу.
[00252] Кроме того, единица-С может дополнительно включать первую вспомогательную часть. Когда единица-С включает первую вспомогательную часть, последовательность первой вспомогательной части расположена в N-концевой области относительно последовательности первого В-клеточного эпитопа в последовательности единицы-С. В частности, первая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции первой вспомогательной части не ограничиваются этим.
[00253] Кроме того, единица-С может дополнительно включать вторую вспомогательную часть. Когда единица-С включает вторую вспомогательную часть, последовательность второй вспомогательной части расположена между последовательностью первого В-клеточного эпитопа и последовательностью первого Th-эпитопа внутри последовательности единицы-С. В частности, вторая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции второй вспомогательной части не ограничиваются этим.
[00254] Кроме того, единица-С может дополнительно включать третью вспомогательную часть. Когда единица-С включает третью вспомогательную часть, последовательность третьей вспомогательной части располагается между последовательностью первого Th-эпитопа и последовательностью второго В-клеточного эпитопа внутри последовательности единицы-С. В частности, третья вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции третьей вспомогательной части не ограничиваются этим.
[00255] Кроме того, единица-С может дополнительно включать четвертую вспомогательную часть. Когда единица-С включает четвертую вспомогательную часть, последовательность четвертой вспомогательной части расположена в C-концевой области относительно последовательности второго В-клеточного эпитопа внутри последовательности единицы-С. В частности, четвертая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции четвертой вспомогательной части не ограничиваются этим.
[00256] структура единицы-C 2 - формула
[00257] В одном варианте осуществления единица-C представляет собой пептид, представленный следующей [формулой C].
[00258][Формула С]
[00259] N-A1-B1-A2-T1-A3-B2-A4-C
[00260] B1 и B2 представляют собой B-клеточные эпитопы, и они следуют принципу конструкции, описанному выше.
[00261] T1 представляет собой Th-эпитоп, и он соответствует принципу конструкции, описанному выше.
[00262] A1, A2, A3 и A4 представляют собой вспомогательные части и могут быть опущены.
[00263] В частности, A1, A2, A3 и A4 могут иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но A1, A2, A3 и A4 не ограничиваются этим.
[00264] длина единицы-C
[00265] В одном варианте осуществления длина единицы-С может соответствовать примерно 24-меру, примерно 25-меру, примерно 26-меру, примерно 27-меру, примерно 28-меру, примерно 29-меру, примерно 30-меру, примерно 31-меру, примерно 32-меру, примерно 33-меру, примерно 34-меру, примерно 35-меру, примерно 36-меру, примерно 37-меру, примерно 38-меру, примерно 39-меру, примерно 40-меру, примерно 41-меру, примерно 42-меру, примерно 43-меру, примерно 44-меру, примерно 45-меру, примерно 46-меру, примерно 47-меру, примерно 48-меру, примерно 49-меру, примерно 50-меру, примерно 51-меру, примерно 52-меру, примерно 53-меру, примерно 54-меру, примерно 55-меру, примерно 56-меру, примерно 57-меру, примерно 58-меру, примерно 59-меру, примерно 60-меру, примерно 61-меру, примерно 62-меру, примерно 63-меру, примерно 64-меру, примерно 65-меру, примерно 66-меру, примерно 67-меру, примерно 68-меру, примерно 69-меру, примерно 70-меру, примерно 71-меру, примерно 72-меру, примерно 73-меру, примерно 74-меру, примерно 75-меру, примерно 76-меру, примерно 77-меру, примерно 78-меру, примерно 79-меру, примерно 80-меру, примерно 81-меру, примерно 82-меру, примерно 83-меру, примерно 84-меру, примерно 85-меру, примерно 86-меру, примерно 87-меру, примерно 88-меру, примерно 89-меру, примерно 90-меру, примерно 91-меру, примерно 92-меру, примерно 93-меру, примерно 94-меру, примерно 95-меру, примерно 96-меру, примерно 97-меру, примерно 98-меру, примерно 99-меру, примерно 100-меру. В другом варианте осуществления длина единицы-C может иметь значение в пределах двух числовых диапазонов, выбранных в непосредственно предшествующем предложении. Например, длина единицы-С может находиться в диапазоне от примерно 24-мера до примерно 45-мера. В другом примере длина единицы-А может находиться в диапазоне от примерно 40-мера до примерно 80-мера.
[00266] вариант осуществления единицы С - примерная конструкция
[00267] В одном варианте осуществления единица-С может иметь последовательность, в которой последовательно связаны первый В-клеточный эпитоп, первая вспомогательная часть, первый Th-эпитоп, вторая вспомогательная часть и второй В-клеточный эпитоп.В частности, каждая из первой вспомогательной части и второй вспомогательной части выполняет линкерную функцию и защитную функцию. Каждая из первой вспомогательной части и второй вспомогательной части включает одну или более искусственных аминокислот.
[00268] В другом варианте осуществления единица-С может иметь последовательность, в которой третья вспомогательная часть, третий В-клеточный эпитоп, четвертая вспомогательная часть, второй Th-эпитоп, пятая вспомогательная часть и четвертый В-клеточный эпитоп последовательно связаны. В частности, третья вспомогательная часть представляет собой His-метку, а каждая из четвертой вспомогательной части и пятой вспомогательной части выполняет линкерную функцию и защитную функцию. Каждая из четвертой вспомогательной части и пятой вспомогательной части включает одну или более искусственных аминокислот.
[00269] варианты осуществления последовательности единицы-C
[00270] В одном варианте осуществления единица-С представляет собой пептидную единицу, выбранную из группы, состоящей из RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF(SEQ ID NO: 107), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLS(SEQ ID NO: 108), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZKTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 109), CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF(SEQ ID NO: 110), CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLS(SEQ ID NO: 31 111), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZKTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 112), KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF (SEQ ID NO: 113), KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLS(SEQ ID NO: 114), KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZKTTKQSFDLSVKAQ YKKNKH(SEQ ID NO: 115), PIFNDVYWIAFK(Cha)VAAWTLKAACRFRGLISLSQ(SEQ ID NO: 116), PPIFNDVYWK(Cha)VAAWTLKAARGLISLSQV(SEQ ID NO: 117), MRGSHHHHHHGSDDDDKIVD RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF(SEQ ID NO: 118), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAF GGGGSGGGGGGSS ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ GGGGSGGGGGGSSCRFRGLISLSQVYLS(SEQ ID NO: 119), RNVPPIFNDVYWIAFILMQYIKANSKFIGICRFRGLISLSQVYLS(SEQ ID NO: 120), RNVPPIFNDVYWIAFZPKYVKQNTLKLATZCRFRGLISLSQVYLS(SEQ ID NO: 121), CRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAFC(SEQ ID NO: 122), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAACRFRGLISLSQVYLS(SEQ ID NO: 160), RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXCRFRGLISLSQVYLS(SEQ ID NO: 198), KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXCRFRGLISLSQVYLS(SEQ ID NO: 201), RNVPPIFNDVYWIAFXPKYVKQNTLKLATXCRFRGLISLSQVYLS(SEQ ID NO: 203), RNVPPIFNDVYWIAFXXKXVAAWTLKAACRFRGLISLSQVYLS(SEQ ID NO: 207), RNVPPIFNDVYWIAFXXKFVAAWTLKAAXXCRFRGLISLSQVYLS(SEQ ID NO: 209), RNVPPIFNDVYWIAFXXKFVAAWTLKAACRFRGLISLSQVYLS(SEQ ID NO: 211), KTTKQSFDLSVKAQYKKNKHZaWPEANQVGAGAFGPGFaZCRFRGLISLSQVY LS(SEQ ID NO: 216), KTTKQSFDLSVKAQYKKNKHZaMDIDPYKEFGATVELLSFLPaZCRFRGLISLS QVYLS(SEQ ID NO: 217), и KTTKQSFDLSVKAQYKKNKHZaILMQYIKANSKFIGIPMGLPQSIALSSLMVAQaZCRFRGLISLSQVYLS(SEQ ID NO: 218). В этом случае «а» обозначает D-форму аланина, «Z» обозначает 6-аминогексановую кислоту, «(Cha)» обозначает L-циклогексилаланин, и «X» обозначает любую стандартную аминокислоту.
[00271] КОНСТРУКЦИЯ ЕДИНИЦЫ-D
[00272] структура единицы-D 1 - обзор
[00273] В качестве пептидной единицы, предложенной в настоящем описании, пептидная единица, 1) которая включает один В-клеточный эпитоп и два Th-эпитопа, 2) в которой последовательность одного Th-эпитопа из двух Th-эпитопов расположена между последовательностью другого Th-эпитопа и последовательностью В-клеточного эпитопа, и 3) которая может включать одну или более вспомогательных частей, называется «единица-С». Функция вспомогательной части конкретно не ограничена, пока она не нарушает функции В-клеточного эпитопа и Th-эпитопа, и при необходимости сконструирована надлежащим образом.
[00274] В одном варианте осуществления единица-D может представлять собой единицу, в которой первый В-клеточный эпитоп, первый Th-эпитоп и второй Th-эпитоп последовательно связаны в направлении от N-конца к С-концу.
[00275] Кроме того, единица-D может дополнительно включать первую вспомогательную часть. Когда единица-D включает первую вспомогательную часть, последовательность первой вспомогательной части расположена в N-концевой области относительно последовательности первого В-клеточного эпитопа в последовательности единицы-D. В частности, первая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции первой вспомогательной части не ограничиваются этим.
[00276] Кроме того, единица-D может дополнительно включать вторую вспомогательную часть. Когда единица-D включает вторую вспомогательную часть, последовательность второй вспомогательной части расположена между последовательностью первого В-клеточного эпитопа и последовательностью первого Th-эпитопа внутри последовательности единицы-D. В частности, вторая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции второй вспомогательной части не ограничиваются этим.
[00277] Кроме того, единица-D может дополнительно включать третью вспомогательную часть. Когда единица-D включает третью вспомогательную часть, последовательность третьей вспомогательной части расположена между последовательностью первого Th-эпитопа и последовательностью второго Th-эпитопа внутри последовательности единицы-D. В частности, третья вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции третьей вспомогательной части не ограничиваются этим.
[00278] Кроме того, единица-D может дополнительно включать четвертую вспомогательную часть. Когда единица-D включает четвертую вспомогательную часть, последовательность четвертой вспомогательной части расположена в C-концевой области относительно последовательности второго Th-эпитопа в последовательности единицы-D. В частности, четвертая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции четвертой вспомогательной части не ограничиваются этим.
[00279] В другом варианте осуществления единица-D может представлять собой единицу, в которой третий Th-эпитоп, четвертый Th-эпитоп и второй В-клеточный эпитоп последовательно связаны в направлении от N-конца к С-концу.
[00280] Кроме того, единица-D может дополнительно включать пятую вспомогательную часть. Когда единица-D включает пятую вспомогательную часть, последовательность пятой вспомогательной части расположена в N-концевой области относительно последовательности третьего Th-эпитопа внутри последовательности единицы-D. В частности, пятая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции пятой вспомогательной части не ограничиваются этим.
[00281] Кроме того, единица-D может дополнительно включать шестую вспомогательную часть. Когда единица-D включает шестую вспомогательную часть, последовательность шестой вспомогательной части располагается между последовательностью третьего Th-эпитопа и последовательностью четвертого Th-эпитопа внутри последовательности единицы-D. В частности, шестая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции шестой вспомогательной части не ограничиваются этим.
[00282] Кроме того, единица-D может дополнительно включать седьмую вспомогательную часть. Когда единица-D включает седьмую вспомогательную часть, последовательность седьмой вспомогательной части расположена между последовательностью четвертого Th-эпитопа и последовательностью второго В-клеточного эпитопа внутри последовательности единицы-D. В частности, седьмая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции седьмой вспомогательной части не ограничиваются этим.
[00283] Кроме того, единица-D может дополнительно включать восьмую вспомогательную часть. Когда единица-D включает восьмую вспомогательную часть, последовательность восьмой вспомогательной части расположена в C-концевой области относительно последовательности второго В-клеточного эпитопа внутри последовательности единицы-D. В частности, восьмая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции восьмой вспомогательной части не ограничиваются этим.
[00284] структура единицы-D 2 - формула
[00285] В одном варианте осуществления единица-В представляет собой пептид, представленный следующей [Формулой D] или [Формулой D'].
[00286][Формула D]
[00287] N-A1-B1-A2-T1-A3-T2-A4-C
[00288][Формула D']
[00289] N-A5-T3-A6-T4-A7-B2-A8-C
[00290] B1 и B2 представляют собой B-клеточные эпитопы, и они следуют принципу конструкции, описанному выше.
[00291] T1-T4 представляют собой Th-эпитопы, и они следуют принципу конструкции, описанному выше.
[00292] A1-A8 представляют собой вспомогательные части и могут быть опущены.
[00293] В частности, A1-A8 могут иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но A1-A8 этим не ограничиваются.
[00294] длина единицы-D
[00295] В одном варианте осуществления длина единицы-D может соответствовать примерно 24-меру, примерно 25-меру, примерно 26-меру, примерно 27-меру, примерно 28-меру, примерно 29-меру, примерно 30-меру, примерно 31-меру, примерно 32-меру, примерно 33-меру, примерно 34-меру, примерно 35-меру, примерно 36-меру, примерно 37-меру, примерно 38-меру, примерно 39-меру, примерно 40-меру, примерно 41-меру, примерно 42-меру, примерно 43-меру, примерно 44-меру, примерно 45-меру, примерно 46-меру, примерно 47-меру, примерно 48-меру, примерно 49-меру, примерно 50-меру, примерно 51-меру, примерно 52-меру, примерно 53-меру, примерно 54-меру, примерно 55-меру, примерно 56-меру, примерно 57-меру, примерно 58-меру, примерно 59-меру, примерно 60-меру, примерно 61-меру, примерно 62-меру, примерно 63-меру, примерно 64-меру, примерно 65-меру, примерно 66-меру, примерно 67-меру, примерно 68-меру, примерно 69-меру, примерно 70-меру, примерно 71-меру, примерно 72-меру, примерно 73-меру, примерно 74-меру, примерно 75-меру, примерно 76-меру, примерно 77-меру, примерно 78-меру, примерно 79-меру, примерно 80-меру, примерно 81-меру, примерно 82-меру, примерно 83-меру, примерно 84-меру, примерно 85-меру, примерно 86-меру, примерно 87-меру, примерно 88-меру, примерно 89-меру, примерно 90-меру, примерно 91-меру, примерно 92-меру, примерно 93-меру, примерно 94-меру, примерно 95-меру, примерно 96-меру, примерно 97-меру, примерно 98-меру, примерно 99-меру, или примерно 100-меру. В другом варианте осуществления длина единицы-В может иметь значение в пределах двух числовых диапазонов, выбранных в непосредственно предшествующем предложении. Например, длина единицы-D может находиться в диапазоне от примерно 24-мера до примерно 45-мера. В другом примере длина единицы-А может находиться в диапазоне от примерно 40-мера до примерно 80-мера.
[00296] вариант осуществления единицы-D - примерная конструкция
[00297] В одном варианте осуществления единица-D может иметь последовательность, в которой последовательно связаны первая вспомогательная часть, первый В-клеточный эпитоп, вторая вспомогательная часть, первый Th-эпитоп, третья вспомогательная часть и второй Th-эпитоп.В частности, первая вспомогательная часть представляет собой His-метку, каждая из второй вспомогательной части и третьей вспомогательной части выполняет линкерную функцию. Каждая из второй вспомогательной части и третьей вспомогательной части включает одну или более искусственных аминокислот.
[00298] В другом варианте осуществления единица-D может иметь последовательность, в которой четвертая вспомогательная часть, второй В-клеточный эпитоп, пятая вспомогательная часть, третий Th-эпитоп, шестая вспомогательная часть, четвертый Th-эпитоп и седьмая вспомогательная часть последовательно связаны. В частности, четвертая вспомогательная часть представляет собой His-метку, каждая из пятой вспомогательной части и шестой вспомогательной части выполняет линкерную функцию, а седьмая вспомогательная часть выполняет защитную функцию. Каждая из пятой вспомогательной части, шестой вспомогательной части и седьмой вспомогательной части включает одну или более искусственных аминокислот.
[00299] Еще в одном варианте осуществления единица-D включает третий В-клеточный эпитоп, восьмую вспомогательную часть, пятый Th-эпитоп, девятую вспомогательную часть и шестой Th-эпитоп.В частности, восьмая вспомогательная часть и девятая вспомогательная часть выполняют линкерную функцию. Каждая из восьмой вспомогательной части и девятой вспомогательной части выполняет защитную функцию, каждая включает одну или более искусственных аминокислот.
[00300] Еще в одном варианте осуществления единица-D включает четвертый В-клеточный эпитоп, десятую вспомогательную часть, седьмой Th-эпитоп, одиннадцатую вспомогательную часть, восьмой Th-эпитоп и двенадцатую вспомогательную часть. В частности, каждая из десятой вспомогательной части и одиннадцатой вспомогательной части выполняет линкерную функцию. Двенадцатая вспомогательная часть имеет защитную функцию. Каждая из десятой вспомогательной части, одиннадцатой вспомогательной части и двенадцатой вспомогательной части включает одну или более искусственных аминокислот.
[00301] варианты осуществления последовательности единицы-D
[00302] В одном варианте осуществления единица-D представляет собой пептидную единицу, выбранную из группы, состоящей из RNVPPIFNDVYWIAF ZaK(Cha)VAAWTLKAAaZ ILMQYIKANSKFIGI(SEQ ID NO: 123), CRFRGLISLSQVYLS ZaK(Cha)VAAWTLKAAaZ ILMQYIKANSKFIGI(SEQ ID NO: 124), KTTKQSFDLSVKAQYKKNKH ZaK(Cha)VAAWTLKAAaZ ILMQYIKANSKFIGI(SEQ ID NO: 125), ILMQYIKANSKFIGI ZaK(Cha)VAAWTLKAAaZ RNVPPIFNDVYWIAF(SEQ ID NO: 126), ILMQYIKANSKFIGI ZaK(Cha)VAAWTLKAAaZ CRFRGLISLSQVYLS(SEQ ID NO: 127), ILMQYIKANSKFIGI ZaK(Cha)VAAWTLKAAaZ KTTKQSFDLSVKAQYKKNKH(SEQ ID NO: 128), PIFNDVYWIAF K(Cha)VAAWTLKAA K(Cha)VAAWTLKAA(SEQ ID NO: 129), PPIFNDVYW K(Cha)VAAWTLKAA K(Cha)VAAWTLKAA(SEQ ID NO: 130), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ ILMQYIKANSKFIGI(SEQ ID NO: 131), MRGSHHHHHHGSDDDDKIVDILMQYIKANSKFIGIZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 132), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGI(SEQ ID NO: 133), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSILMQ YIKANSKFIGIPMGLPQSIALSSLMVAQGGGGSGGGGGGSSILMQYIKANSKFI GIPMGLPQSIALSSLMVAQ(SEQ ID NO: 134), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZK(Cha)LAAFTIRAAaZ(SEQ ID NO: 135), и CRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIC(SEQ ID NO: 136). В этом случае «а» обозначает D-форму аланина, «Z» обозначает 6-аминогексановую кислоту, «(Cha)» обозначает L-циклогексилаланин, и «X» обозначает любую стандартную аминокислоту.
[00303] КОНСТРУКЦИЯ ЕДИНИЦЫ-E
[00304] структура единицы-Е 1 - обзор
[00305] В качестве пептидной единицы, предложенной в настоящем описании, пептидная единица 1), которая включает два В-клеточных эпитопа и два Th-эпитопа, 2) в которой каждая последовательность двух Th-эпитопов расположена между последовательностью одного В-клеточного эпитопа из двух В-клеточных эпитопов и последовательностью другого В-клеточного эпитопа, и 3) которая может включать одну или более вспомогательных частей, называется «единицей-Е». Функция вспомогательной части конкретно не ограничена, пока она не нарушает функции В-клеточного эпитопа и Th-эпитопа, и при необходимости сконструирована надлежащим образом.
[00306] В одном варианте осуществления единица-Е может представлять собой единицу, в которой первый В-клеточный эпитоп, первый Th-эпитоп, второй Th-эпитоп и второй В-клеточный эпитоп последовательно связаны в направлении от N-конца к С-концу.
[00307] Кроме того, единица-Е может дополнительно включать первую вспомогательную часть. Когда единица-Е включает первую вспомогательную часть, последовательность первой вспомогательной части расположена в N-концевой области относительно последовательности первого В-клеточного эпитопа в последовательности единицы-Е. В частности, первая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции первой вспомогательной части не ограничиваются этим.
[00308] Кроме того, единица-Е может дополнительно включать вторую вспомогательную часть. Когда единица-Е включает вторую вспомогательную часть, последовательность второй вспомогательной части расположена между последовательностью первого В-клеточного эпитопа и последовательностью первого Th-эпитопа внутри последовательности единицы-Е. В частности, вторая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции второй вспомогательной части не ограничиваются этим.
[00309] Кроме того, единица-Е может дополнительно включать третью вспомогательную часть. Когда единица-Е включает третью вспомогательную часть, последовательность третьей вспомогательной части расположена между последовательностью первого Th-эпитопа и последовательностью второго Th-эпитопа внутри последовательности единицы-Е. В частности, третья вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции третьей вспомогательной части не ограничиваются этим.
[00310] Кроме того, единица-Е может дополнительно включать четвертую вспомогательную часть. Когда единица-Е включает четвертую вспомогательную часть, последовательность четвертой вспомогательной части расположена между последовательностью второго В-клеточного эпитопа и второго В-клеточного эпитопа внутри последовательности единицы-Е. В частности, четвертая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или защитную функцию, но функции четвертой вспомогательной части не ограничиваются этим.
[00311] Кроме того, единица-Е может дополнительно включать пятую вспомогательную часть. Когда единица-Е включает пятую вспомогательную часть, последовательность пятой вспомогательной части расположена в N-концевой области относительно последовательности второго В-клеточного эпитопа в последовательности единицы-Е. В частности, пятая вспомогательная часть может иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но функции пятой вспомогательной части не ограничиваются этим.
[00312] структура единицы-Е 2 - формула
[00313] В одном варианте осуществления единица-Е представляет собой пептид, представленный следующей [Формулой Е].
[00314] [Формула Е]
[00315] N-A1-B1-A2-T1-A3-T2-A4-B2-A5-C
[00316] B1 и B2 представляют собой B-клеточные эпитопы, и они следуют принципу конструкции, описанному выше.
[00317] T1 и T2 представляют собой Th-эпитопы, и они следуют принципу конструкции, описанному выше.
[00318] A1, A2, A3, A4 и A5 представляют собой вспомогательные части и могут быть опущены.
[00319] В частности, A1, A2, A3, A4 и A5 могут иметь фиктивную функцию, функцию повышения растворимости, линкерную функцию и/или функцию образования циклической формы, но A1, A2, A3, A4 и A5 этим не ограничиваются.
[00320] длина единицы-E
[00321] В одном варианте осуществления длина единица-Е может соответствовать примерно 32-меру, примерно 33-меру, примерно 34-меру, примерно 35-меру, примерно 36-меру, примерно 37-меру, примерно 38-меру, примерно 39-меру, примерно 40-меру, примерно 41-меру, примерно 42-меру, примерно 43-меру, примерно 44-меру, примерно 45-меру, примерно 46-меру, примерно 47-меру, примерно 48-меру, примерно 49-меру, примерно 50-меру, примерно 51-меру, примерно 52-меру, примерно 53-меру, примерно 54-меру, примерно 55-меру, примерно 56-меру, примерно 57-меру, примерно 58-меру, примерно 59-меру, примерно 60-меру, примерно 61-меру, примерно 62-меру, примерно 63-меру, примерно 64-меру, примерно 65-меру, примерно 66-меру, примерно 67-меру, примерно 68-меру, примерно 69-меру, примерно 70-меру, примерно 71-меру, примерно 72-меру, примерно 73-меру, примерно 74-меру, примерно 75-меру, примерно 76-меру, примерно 77-меру, примерно 78-меру, примерно 79-меру, примерно 80-меру, примерно 81-меру, примерно 82-меру, примерно 83-меру, примерно 84-меру, примерно 85-меру, примерно 86-меру, примерно 87-меру, примерно 88-меру, примерно 89-меру, примерно 90-меру, примерно 91-меру, примерно 92-меру, примерно 93-меру, примерно 94-меру, примерно 95-меру, примерно 96-меру, примерно 97-меру, примерно 98-меру, примерно 99-меру, или примерно 100-меру. В другом варианте осуществления длина единицы-Е может иметь значение в пределах двух числовых диапазонов, выбранных в непосредственно предшествующем предложении. Например, длина единицы-Е может находиться в диапазоне от примерно 32-мера до примерно 60-мера. В другом примере длина единицы-А может находиться в диапазоне от примерно 50-мера до примерно 100-мера.
[00322] вариант осуществления единицы-Е - примерная конструкция
[00323] В одном варианте осуществления единица-Е может иметь последовательность, в которой первая вспомогательная часть, первый В-клеточный эпитоп, вторая вспомогательная часть, первый Th-эпитоп, третья вспомогательная часть, второй Th-эпитоп, четвертая вспомогательная часть, и второй В-клеточный эпитоп последовательно связаны. В частности, первой вспомогательной частью является His-метка. Каждая из второй вспомогательной части и четвертой вспомогательной части выполняет линкерную функцию и защитную функцию. Третья вспомогательная часть выполняет линкерную функцию. Вторая вспомогательная часть, третья вспомогательная часть и четвертая вспомогательная часть включают одну или более искусственных аминокислот.
[00324] В другом варианте осуществления единица-Е может иметь последовательность, в которой третий В-клеточный эпитоп, пятая вспомогательная часть, третий Th-эпитоп, шестая вспомогательная часть, четвертый Th-эпитоп, седьмая вспомогательная часть и четвертый В-клеточный эпитоп последовательно связаны. В частности, каждая из пятой вспомогательной части и седьмой вспомогательной части выполняет линкерную функцию и защитную функцию. Шестая вспомогательная часть выполняет линкерную функцию. Пятая вспомогательная часть, шестая вспомогательная часть и седьмая вспомогательная часть включают одну или более искусственных аминокислот.
[00325] варианты осуществления последовательности единицы-E
[00326] В одном варианте осуществления единица-Е представляет собой пептидную единицу, выбранную из группы, состоящей из RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIRNVPPIFN DVYWIAF(SEQ ID NO: 137), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGICRFRGLIS LSQVYLS(SEQ ID NO: 138), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIKTTKQSF DLSVKAQYKKNKH(SEQ ID NO: 139), CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIRNVPPIFN DVYWIAF(SEQ ID NO: 140), CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGICRFRGLIS LSQVYLS(SEQ ID NO: 141), CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIKTTKQSF DLSVKAQYKKNKH(SEQ ID NO: 142), KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIR NVPPIFNDVYWIAF(SEQ ID NO: 143), KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIC RFRGLISLSQVYLS(SEQ ID NO: 144), KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIK TTKQSFDLSVKAQYKKNKH(SEQ ID NO: 145), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ ILMQYIKANSKFIGIRNVPPIFNDVYWIAF(SEQ ID NO: 146), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSILMQ YIKANSKFIGIPMGLPQSIALSSLMVAQILMQYIKANSKFIGIPMGLPQSIALSSL MVAQGGGGSGGGGGGSSCRFRGLISLSQVYLS(SEQ ID NO: 147), PIFNDVYWIAFK(Cha)VAAWTLKAAK(Cha)VAAWTLKAACRFRGLISLSQ(SEQ ID NO: 148), PPIFNDVYWK(Cha)VAAWTLKAAK(Cha)VAAWTLKAARGLISLSQV(SEQ ID NO: 149), и CRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIRNVPPIF NDVYWIAFC(SEQ ID NO: 150). В этом случае «а» обозначает D-форму аланина, «Z» обозначает 6-аминогексановую кислоту, «(Cha)» обозначает L-циклогексилаланин.
[00327] КОНСТРУКЦИЯ ПЕПТИДА
[00328] конструкция пептида - обзор
[00329] Предложенные в настоящем описании пептиды можно сконструировать с использованием одной или более пептидных единиц, описанных выше. Эти пептиды включают одну или более пептидных единиц и могут включать один или более типов пептидных единиц. Например, пептид может быть сконструирован путем 1) включения только одной пептидной единицы, 2) конструирования конкатемера путем связывания нескольких пептидных единиц одного типа, имеющих одинаковую последовательность, 3) конструирования пептида в виде нитки бус с помощью связывание одного или более типов пептидных единиц с различными последовательностями, 4) путем смешивания способов конструирования 1) и 3), указанных выше, и 5) конструирования пептида в циклической форме путем соединения обоих концов пептида, сконструированного указанным выше способом, но способы конструирования не ограничиваются этим. Далее каждый метод конструирования будет описан подробно.
[00330] конструкция одной единицы
[00331] Пептид может быть сконструирован так, чтобы включать только одну из пептидных единиц, описанных выше. В одном варианте осуществления пептид может включать одну пептидную единицу, выбранную из группы, состоящей из единицы-А, единицы-В, единицы-С, единицы-D и единицы-Е. В частности, пептидная единица имеет строение, описанное выше.
[00332] конструкция конкатамера 1 - обзор
[00333] Пептид может быть сконструирован в виде конкатемера, в котором соединены несколько пептидных единиц, имеющих одинаковую последовательность. Пептид, сконструированный в виде конкатемера, состоит из 1) одного типа пептидной единицы и 2) нескольких пептидных единиц, имеющих одинаковую или эквивалентную последовательность.
[00334] В частности, две пептидные единицы с «эквивалентной последовательностью» относятся к случаям, когда 1) вспомогательная часть присутствует на N-конце и/или С-конце каждого из двух пептидов и 2) когда вспомогательная часть присутствует, даже если две последовательности вспомогательных частей различаются, остальная часть последовательности такая же. Например, когда первый пептид имеет последовательность, в которой первая вспомогательная часть и первая единица-А связаны в направлении от N-конца к С-концу, второй пептид имеет последовательность, в которой первая единица-А и первая вспомогательная часть связаны в направлении от N-конца к С-концу, третий пептид имеет последовательность, в которой вторая вспомогательная часть, первая единица-А и третья вспомогательная часть связаны в направлении от N-конца к С-концу, и четвертый пептид имеет первую последовательность единицы-А, говорят, что пептиды с первого по четвертый имеют эквивалентную последовательность.
[00335] В одном варианте осуществления пептид может включать пептид, в котором первая пептидная единица и вторая пептидная единица соединены по порядку. В частности, первая пептидная единица представляет собой пептидную единицу, выбранную из группы, состоящей из единицы-А, единицы-В, единицы-С, единицы-D и единицы-Е; и вторая пептидная единица имеет последовательность, которая является такой же или эквивалентной первой пептидной единице.
[00336] В другом варианте осуществления пептид может включать пептид, в котором третья пептидная единица, четвертая пептидная единица и пятая пептидная единица связаны по порядку. В частности, третья пептидная единица представляет собой пептидную единицу, выбранную из группы, состоящей из единицы-А, единицы-В, единицы-С, единицы-D и единицы-Е; и каждая из четвертой пептидной единицы и пятой пептидной единицы имеет последовательность, которая является такой же или эквивалентной третьей пептидной единице.
[00337] Еще в одном варианте осуществления пептид может включать пептид, в котором шестая пептидная единица, седьмая пептидная единица, восьмая пептидная единица и девятая пептидная единица соединены по порядку. В частности, третья пептидная единица представляет собой пептидную единицу, выбранную из группы, состоящей из единицы-А, единицы-В, единицы-С, единицы-D и единицы-Е; и седьмая пептидная единица, восьмая пептидная единица и девятая пептидная единица имеют последовательность, которая является такой же или эквивалентной шестой пептидной единице.
[00338] конструкция конкатемера 2 - формула
[00339] В одном варианте осуществления пептид может представлять собой пептид, представленный следующей [Формулой 1].
[00340][Формула 1]
[00341] N-U1-U2-…-Un-C
[00342] где U1 - Un каждая представляет собой пептидную единицу, выбранную из группы, состоящей из единицы-А, единицы-В, единицы-С, единицы-D и единицы-Е, и они имеют состав пептидных единиц, описанный выше.
[00343] U1 - Un имеют одинаковую или эквивалентную последовательность.
[00344] n представляет собой целое число, равное 2 или больше.
[00345] конструкция конкатемера 3 - примерные последовательности
[00346] В одном варианте осуществления пептид представляет собой пептид, выбранный из группы, состоящей из MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 151), RNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZRNVPPIF NDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ(SEQ ID NO: 152), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLSRNVPPIF NDVYWIAFZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLS(SEQ ID NO: 153), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIRNVPPIFN DVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGI(SEQ ID NO: 154), и RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIRNVPPIFN DVYWIAFRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGI RNVPPIFNDVYWIAF(SEQ ID NO: 155). В этом случае «а» обозначает D-форму аланина, «Z» обозначает 6-аминогексановую кислоту, «(Cha)» обозначает L-циклогексилаланин.
[00347] конструкция нитка бус 1 - обзор
[00348] Пептид может быть сконструирован в виде нитки бус, в которой соединены несколько пептидных единиц, имеющих разные последовательности. Пептид, сконструированный в виде нитки бус, состоит из 1) по меньшей мере одного вида пептидной единицы и 2) множества пептидных единиц, имеющих разные последовательности.
[00349] В одном варианте осуществления пептид может включать пептид, в котором первая пептидная единица и вторая пептидная единица последовательно связаны. В частности, первая пептидная единица и вторая пептидная единица, каждая, представляет собой пептидную единицу, выбранную из группы, состоящей из единицы-А, единицы-В, единицы-С, единицы-D и единицы-Е; и первая пептидная единица и вторая пептидная единица имеют последовательности, отличающиеся друг от друга.
[00350] В другом варианте осуществления пептид может включать пептид, в котором последовательно связаны третья пептидная единица, четвертая пептидная единица и пятая пептидная единица. В частности, каждая из третьей пептидной единицы, четвертой пептидной единицы и пятой пептидной единицы представляет собой пептидную единицу, выбранную из группы, состоящей из единицы-А, единицы-В, единицы-С, единицы-D и единицы-Е; и третья пептидная единица, четвертая пептидная единица и пятая пептидная единица имеют последовательности, отличающиеся друг от друга.
[00351] Еще в одном варианте осуществления пептид может включать пептид, в котором последовательно связаны шестая пептидная единица, седьмая пептидная единица, восьмая пептидная единица и девятая пептидная единица. В частности, шестое пептидная единица, седьмое пептидная единица, восьмое пептидная единица и девятое пептидная единица, каждое, представляет собой пептидная единица, выбранное из группы, состоящей из единицы-А, единицы-В, единицы-С, единицы-D, и единица-Е; и шестая пептидная единица, седьмая пептидная единица, восьмая пептидная единица и девятая пептидная единица имеют последовательности, отличающиеся друг от друга.
[00352] конструкция нитка бус 2 - обзор
[00353] В одном варианте осуществления пептид может представлять собой пептид, представленный следующей [Формулой 2].
[00354][Формула 2]
[00355] N-U1-U2-…-Un-C
[00356] где U1 - Un каждая представляет собой пептидную единицу, выбранную из группы, состоящей из единицы-А, единицы-В, единицы-С, единицы-D и единицы-Е, и они имеют состав пептидных единиц, описанный выше.
[00357] U1 - Un имеют одинаковую или эквивалентную последовательность.
[00358] n представляет собой целое число, равное 2 или больше.
[00359] конструкция нитка бус 3 - примерные последовательности
[00360] В одном варианте осуществления пептид может представлять собой пептид RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAFCRFRGLI SLSQVYLSZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 156), в котором «a» обозначает D-форму аланина, Z обозначает 6-аминогексановую кислоту и (Cha) обозначает L-циклогексилаланин.
[00361] смешанная конструкция 1 - обзор
[00362] Пептид может быть сконструирован путем соответствующего смешивания вышеупомянутых 1) конструкции одной единицы, 2) конструкции конкатемера и 3) конструкции в виде нитки бус.В одном варианте осуществления пептид может быть сконструирован путем сначала конструирования пептидной единицы в соответствии со способами конструирования, описанными выше, а затем связывания множества пептидных единиц.
[00363] В одном варианте осуществления пептид может представлять собой пептид, в котором последовательно связаны первая пептидная единица и вторая пептидная единица. В частности, первая пептидная единица и вторая пептидная единица, каждая, имеет структуру пептида в соответствии с любой из конструкций одной единицы, конструкции конкатемера и конструкции в виде нитки бус, а также последовательности первой пептидной единицы и последовательности второй пептидной единицы отличаются друг от друга.
[00364] В другом варианте осуществления пептид может представлять собой пептид, в котором последовательно связаны третья пептидная единица, четвертая пептидная единица и пятая пептидная единица. В частности, третья пептидная единица, четвертая пептидная единица и пятая пептидная единица, каждая, имеет структуру пептида в соответствии с любой из конструкции одной единицы, конструкции конкатемера и конструкции в виде нитки бус, а также последовательности третьей пептидной единицы, четвертой пептидной единицы и пятой пептидной единицы отличаются друг от друга.
[00365] Еще в одном варианте осуществления пептид может представлять собой пептид, в котором шестая пептидная единица, седьмая пептидная единица, восьмая пептидная единица и девятая пептидная единица соединены последовательно. В частности, шестая пептидная единица, седьмая пептидная единица, восьмая пептидная единица и девятая пептидная единица имеют структуру пептида в соответствии с любой из конструкций одной единицы, конструкции конкатемера и конструкции в виде нитки бус, и последовательности шестой пептидной единицы, седьмой пептидной единицы, восьмой пептидной единицы и девятой пептидной единицы отличаются друг от друга.
[00366] смешанная конструкция 2 - формула
[00367] В одном варианте осуществления пептид может представлять собой пептид, представленный следующей [Формулой 3]:
[00368][Формула 3]
[00369] N-P1-P2-…-Pn-C
[00370] в которой P1 - Pn каждый представляет собой пептидную единицу, сконструированную методом, выбранным из группы, состоящей из конструкции одной единицы, конструкции конкатемера и конструкции в виде нитки бус, и они имеют конструкции и составы пептидов, описанные выше.
[00371] n представляет собой целое число, равное 2 или больше.
[00372] смешанная конструкция 3 - примерные последовательности
[00373] В одном варианте осуществления пептид представляет собой пептид, выбранный из группы, состоящей из RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAFZRNVPPI FNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIZRNVPPIFNDVY WIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGICRFRGLISLSQVYLS(SEQ ID NO: 157), и RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAFCRFRGLI SLSQVYLSZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAFZRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIZRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ ILMQYIKANSKFIGICRFRGLISLSQVYLS(SEQ ID NO: 158). В этом случае «а» обозначает D-форму аланина, «Z» обозначает 6-аминогексановую кислоту, а «(Cha)» обозначает L-циклогексилаланин.
[00374] конструкция циклической формы
[00375] Пептид может быть сконструирован так, чтобы образовывать циклическую форму. Когда пептид находится в циклической форме, повышается стабильность в организме пациента; поэтому можно ожидать улучшенного эффекта при использовании пептида с циклической формой в качестве иммунотерапевтического средства. В одном варианте осуществления, что касается пептида, сконструированного с помощью метода конструирования, выбранного из группы, состоящей из конструкции одной единицы, конструкции конкатемера, конструкции в виде нитки бус и метода смешанной конструкции, пептид может быть сконструирован так, чтобы дополнительно иметь вспомогательную часть, выполняющую функцию образования циклической формы на N-конце и С-конце. В другом варианте осуществления, что касается пептида, сконструированного с помощью метода конструкции, выбранного из группы, состоящей из конструкции одной единицы, конструкции конкатемера, конструкции в виде нитки бус и метода смешанной конструкции, пептид может быть сконструирован так, чтобы дополнительно включать вспомогательную часть и образовывать циклическую форму через вспомогательную часть.
[00376] другие конструкции
[00377] Пептид может быть сконструирован другими способами, если это необходимо, в дополнение к методам конструирования, описанным выше. В одном варианте осуществления, что касается пептида, сконструированного с помощью метода конструирования, выбранного из группы, состоящей из конструкции одной единицы, конструкции конкатемера, конструкции в виде нитки бус и метода смешанной конструкции, пептид может дополнительно включать одну или более вспомогательных частей, один или более В-клеточных эпитопов и/или один или более Th-эпитопов.
[00378] последовательности, сходные с раскрытыми пептидными единицами и/или пептидами
[00379] В настоящем описании пептидные единицы и/или пептиды, которые имеют последовательность, подобную той, что раскрыта в разделах «конструкция единицы-А», «конструкция единицы-В», «конструкция единицы-С», «конструкция единицы-D», «конструкция единицы-Е» и «конструкция пептида», раскрыты выше.
[00380] В одном варианте осуществления пептидная единица может иметь последовательность, имеющую 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с любой из последовательностей, раскрытых в разделах «конструкция единицы-А», «конструкция единицы-В», «конструкция единицы-С», «конструкция единицы-D» и «конструкция единицы-Е». В другом варианте осуществления пептидная единица может иметь последовательность, совпадающую с любой из последовательностей, раскрытых в разделах «конструкция единицы-А», «конструкция единицы-В», «конструкция единицы-С», «конструкция единицы-D» и «конструкция единицы-Е» по меньшей мере числовым значением, выбранным в непосредственно предшествующем предложении. Еще в одном варианте осуществления пептидная единица может иметь последовательность, имеющую 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с любой последовательностью, выбранной из SEQ ID NO: 56 - SEQ ID NO: 150, SEQ ID NO: 160 - SEQ ID NO: 161 и SEQ ID NO: 198 - SEQ ID NO: 220. Еще в одном варианте осуществления пептидная единица может иметь последовательность, совпадающую с любой из раскрытых последовательностей, выбранных из последовательностей SEQ ID NO: 56 - SEQ ID NO: 150, SEQ ID NO: 160 - SEQ ID NO: 161 и SEQ ID NO: NO: 198 - SEQ ID NO: 220, по меньшей мере, на числовое значение, выбранное в непосредственно предшествующем предложении. Например, пептидная единица может иметь последовательность, которая имеет 90% или более идентичности с SEQ ID NO: 56.
[00381] Еще в одном варианте осуществления пептид может иметь последовательность, имеющую 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с любой из последовательностей, раскрытых в разделе «конструкция пептида». Еще в одном варианте осуществления пептид может иметь последовательность, совпадающую с любой из последовательностей, описанных в разделе «конструкция пептида», по меньшей мере на числовое значение, выбранное в непосредственно предшествующем предложении. Еще в одном варианте осуществления пептид может иметь последовательность, имеющую 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичности с любой из последовательностей, выбранных из SEQ ID NO: 151 - SEQ ID NO: 158. Еще в одном варианте осуществления пептид может иметь последовательность, совпадающую с любой из последовательностей SEQ ID NO: 151 - SEQ ID NO: 158 по меньшей мере на числовое значение, выбранное в непосредственно предшествующем предложении. Например, пептид может иметь последовательность, которая имеет 90% или более идентичности с SEQ ID NO: 151.
[00382] применение пептидов
[00383] применение пептидов - обзор
[00384] Предложенные в настоящем описании пептиды подходят в качестве иммунотерапевтических средств, поскольку при введении в организм пациента они обладают следующими характеристиками: 1) они индуцируют продукцию антител, которые специфически связываются со специально сконструированными В-клеточными эпитопами, и 2) они индуцируют продукцию однородных антител. Следовательно, пептиды можно использовать в качестве иммунотерапевтических средств. В одном варианте осуществления пептиды, предложенные в настоящем описании, можно использовать в качестве иммунотерапевтического средства для лечения ожирения. В другом варианте осуществления пептидные единицы и/или пептиды, включая те, что предложены в настоящем описании, можно использовать для лечения ожирения.
[00385] Применение пептидов для лечения ожирения
[00386] Предложенные в настоящем описании пептиды включают В-клеточный эпитоп.В одном варианте осуществления В-клеточный эпитоп может представлять собой В-клеточный эпитоп, включенный в SEQ ID NO: 41-75. В частности, известно, что В-клеточный эпитоп индуцирует антитела, обладающие способностью связываться с ApoB-100 (заявка на патент США №10/378,707, PCT/KR2005/000784, и Kim et al., 2016, An apolipoprotein B100 mimotope prevents obesity in mice, Clinical Science 130, 105-116). Из предшествующих документов, приведенных выше, известно, что когда антитело, обладающее способностью связываться с ApoB-100, индуцируется В-клеточным эпитопом в организме пациента, оно оказывает иммунотерапевтическое действие на ожирение. Соответственно, в настоящем описании раскрыты применения пептидов для лечения ожирения и способ применения. Чтобы описать иммунотерапевтические эффекты пептидов при ожирении, заявка на патент США №10/378,707, PCT/KR2005/000784, и Kim et al., 2016, An apolipoprotein B100 mimotope prevents obesity in mice, Clinical Science 130, 105-116 включены в настоящее описание в качестве ссылки. В случае противоречия между частью, на которую делается ссылка, и настоящим описанием следует понимать, что настоящей описание имеет приоритет над частью, на которую делается ссылка.
[00387] фармацевтическая композиция, включающая пептид
[00388] Настоящее описание раскрывает фармацевтическую композицию, включающую описанный выше пептид. Пептид может быть использован в качестве иммунотерапевтического средства и имеет сходство с вакцинами в том, что он индуцирует гуморальный иммунитет при введении в организм. Следовательно, специалисты в данной области могут включить в фармацевтическую композицию, включающую пептид, соответствующий состав, который можно добавлять для введения обычных вакцин и/или для усиления эффекта индукции иммунного ответа. Например, фармацевтическая композиция может включать готовый к применению пептид, фармацевтически приемлемые носители, добавки и/или адъюванты, но не ограничивается ими. В частности, фармацевтическая композиция может включать воду, физиологический раствор, декстрозу, этанол, глицерин, хлорид натрия, декстрозу, маннит, сорбит, лактозу, желатин, альбумин, гидроксид алюминия, неполный адъювант Фрейнда и полный адъювант (Pifco Laboratories, Детройт, Мичиган), адъювант Merck 65 (Merck and Company, Inc., Рауэй, Нью-Джерси), гель гидроксида алюминия (Alum) или соли алюминия, такие как фосфат алюминия, серия AS04, MF, сквален, MF59, QS21, соли кальция, железа или цинка, нерастворимые суспензии ацилированного тирозина, ацилированной фруктозы, полисахариды катионного или анионного происхождения, полифосфазены, биоразлагаемые микросферы, Quil A, агонисты toll-подобного рецептора (TLR), PHAD[Avanti полярный липид, монофосфорил липид A (синтетический)], монофосфорил липид А (MPL, монофосфорил липид А), синтетический липид А, имитаторы или аналоги липида А, соли алюминия, цитокины, сапонины, пролактин, гормона роста, дезоксихолевую кислоту, бетаглюкан, полирибонуклеотиды, производные мурамилдипептида (MDP), олигонуклеотиды CpG, липополисахарид грамотрицательных бактерий (LPS), полифосфазен, эмульсию, виросому, кохлеат, поли(лактид-со-гликолид) (PLG) микрочастицы, полоксамерные частицы, микрочастицы, липосомы или их подходящую комбинацию.
[00389] СПОСОБ ПРОИЗВОДСТВА ПЕПТИДОВ
[00390] Предлагаемые в настоящем описании пептиды могут быть получены известным способом, который может быть использован специалистами в данной области, и способ получения конкретно не ограничен. В одном варианте осуществления пептид может быть получен способом получения рекомбинантного белка. В другом варианте осуществления пептид может быть синтезирован химическим путем. В частности, пептид может быть синтезирован методом жидкофазного синтеза пептида, методом твердофазного синтеза пептида или конвергентным методом малых пептидных фрагментов, но способы не ограничиваются этим.
[00391] нуклеиновая кислота, кодирующая пептидную единицу и/или пептид
[00392] нуклеиновая кислота, кодирующая пептидную единицу и/или пептид - обзор
[00393] В настоящем описании раскрыты нуклеиновые кислоты, которые кодируют пептидные единицы и/или пептиды, раскрытые выше (далее «кодирующая нуклеиновая кислота»). Хотя пептидные единицы и пептиды, раскрытые в настоящем описании, могут включать нестандартные аминокислоты, в природе не существует кодонов, соответствующих нестандартным аминокислотам. Следовательно, нестандартные аминокислоты не могут быть закодированы общим методом. Соответственно, необходимо заменить эти нестандартные аминокислоты соответствующими стандартными аминокислотами, чтобы закодировать их в виде нуклеиновых кислот.Когда пептидная единица и/или пептид не включают нестандартную аминокислоту, кодирующая нуклеиновая кислота может быть сконструирована с использованием кодона нуклеиновой кислоты, соответствующего каждой стандартной аминокислоте.
[00394] Для удобства описания в настоящем описании пептидная единица и/или пептид включает замену нестандартной аминокислоты на подходящую стандартную аминокислоту; и пептидную единицу и/или пептид, включающий только стандартные аминокислоты, называют целевым пептидом, подлежащим кодированию, а ДНК и/или РНК, кодирующие целевой пептид, подлежащий кодированию, называют кодирующей нуклеиновой кислотой. В частности, когда пептидная единица и/или пептид не включает нестандартную аминокислоту, пептидная единица и/или пептид имеет ту же аминокислотную последовательность, что и кодируемый целевой пептид.
[00395] Используемый в настоящем описании термин «кодируемый целевой пептид» представляет собой концептуальный термин, введенный для упрощения описания полученной кодирующей нуклеиновой кислоты, и он не зависит от способа или процедуры получения кодирующей нуклеиновой кислоты.
[00396] конструкция подлежащего кодированию целевого пептида
[00397] Когда пептидная единица и пептид, описанные в настоящем документе, включают нестандартную аминокислоту, кодируемый целевой пептид создается путем замены его соответствующей стандартной аминокислотой. Когда пептидная единица и пептид, раскрытые в настоящем описании, не включают нестандартную аминокислоту, соответствующий кодируемый целевой пептид имеет ту же последовательность, что и пептидная единица и пептид. В одном варианте осуществления кодируемый целевой пептид может представлять собой пептид, в котором нестандартная аминокислота заменена любой стандартной аминокислотой. В другом варианте осуществления кодируемый целевой пептид может представлять собой пептид, в котором нестандартная аминокислота заменена стандартной аминокислотой, идентичной ей или имеющей эквивалентную функцию. Еще в одном варианте осуществления кодируемый целевой пептид может иметь ту же последовательность, что и пептидная единица и/или пептид. В частности, пептидная единица и/или пептид характеризуются отсутствием нестандартных аминокислот.
[00398] варианты осуществления конструкции целевого пептида, подлежащего кодированию - случаи, включающие PADRE
[00399] Пептидная единица и пептид, описанные в настоящем документе, включают один или более Th-эпитопов. В частности, когда Th-эпитоп представляет собой последовательность, названную PADRE, описанную в заявке на патент США №305871, она может включать нестандартную аминокислоту, L-циклогексилаланин. Согласно литературным данным, L-циклогексилаланин может иметь как функцию защиты PADRE от фермента, разрушающего пептид, так и функцию якорного остатка, способного связываться с МНС класса II. Таким образом, целевой пептид, который подлежит кодированию, конструируют путем замены L-циклогексилаланина на подходящую стандартную аминокислоту, обладающую такой же или эквивалентной функцией. В одном варианте осуществления L-циклогексилаланин может быть заменен любой стандартной аминокислотой. В другом варианте осуществления L-циклогексилаланин может быть заменен фенилаланином или тирозином. Еще в одном варианте осуществления последовательность целевого пептида, который подлежит кодированию в соответствии с PADRE, может представлять собой KFVAAWTLKAA (SEQ ID NO: 195), KYVAAWTLKAA (SEQ ID NO: 196), или KXVAAWTLKAA (SEQ ID NO: 197), в которой X относится к любой стандартной аминокислоте.
[00400] варианты последовательности целевого пептида, который подлежит кодированию
[00401] В одном варианте осуществления последовательность целевого пептида, подлежащего кодированию, выбирают из RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXCRFRGLISLSQVYLS(SEQ ID NO: 198), RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXGSHHHHHHGSDDDDK(SEQ ID NO: 199), GSHHHHHHGSDDDDKXXKXVAAWTLKAAXXRNVPPIFNDVYWIAF(SEQ ID NO: 200), KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXCRFRGLISLSQVYLS(SEQ ID NO: 201), RNVPPIFNDVYWIAFCRFRGLISLSQVYLSXXK(Cha)VAAWTLKAAXX(SEQ ID NO: 202), RNVPPIFNDVYWIAFXPKYVKQNTLKLATXCRFRGLISLSQVYLS(SEQ ID NO: 203), RNVPPIFNDVYWIAFXXKXVAAWTLKAAXX(SEQ ID NO: 204), RNVPPIFNDVYWIAFKXVAAWTLKAA(SEQ ID NO: 205), RNVPPIFNDVYWIAFKXVAAWTLKAAHHHHHH(SEQ ID NO: 206), RNVPPIFNDVYWIAFXXKXVAAWTLKAACRFRGLISLSQVYLS(SEQ ID NO: 207), RNVPPIFNDVYWIAFXXKXVAAWTLKAACR(SEQ ID NO: 208), RNVPPIFNDVYWIAFXXKFVAAWTLKAAXXCRFRGLISLSQVYLS(SEQ ID NO: 209), RNVPPIFNDVYWIAFXXKFVAAWTLKAAXX(SEQ ID NO: 210), RNVPPIFNDVYWIAFXXKFVAAWTLKAACRFRGLISLSQVYLS(SEQ ID NO: 211), и RNVPPIFNDVYWIAFXXKFVAAWTLKAACR(SEQ ID NO: 212). В этом случае «X» обозначает любую стандартную аминокислоту.
[00402] конструкция кодирующей нуклеиновой кислоты 1 - на основе кодонов нуклеиновой кислоты
[00403] Раскрытая в настоящем описании кодирующая нуклеиновая кислота относится к кодонам нуклеиновой кислоты, кодирующим целевой пептид. Поскольку все последовательности кодируемых целевых пептидов представляют собой стандартные аминокислоты, кодирующую нуклеиновую кислоту конструируют на основе кодонов нуклеиновой кислоты, соответствующих каждой аминокислоте кодируемого целевого пептида. В частности, поскольку один или более кодонов нуклеиновой кислоты могут соответствовать одной стандартной аминокислоте, в конечном итоге могут быть сконструированы две или более кодирующие нуклеиновые кислоты, кодирующие один целевой пептид, который подлежит кодированию. В одном варианте осуществления кодирующая нуклеиновая кислота может представлять собой кодоны ДНК и/или РНК, кодирующие подлежащий кодированию целевой пептид. В другом варианте осуществления кодирующая нуклеиновая кислота может иметь последовательность ДНК и/или РНК, которая способна к комплементарному связыванию с кодонами ДНК и/или РНК, кодирующими подлежащий кодированию целевой пептид.
[00404] Последовательность кодирующей нуклеиновой кислоты может быть оптимизирована по кодонам, что более подробно описано ниже.
[00405] конструкция кодирующей нуклеиновой кислоты 2 - оптимизация по кодонам
[00406] Как описано выше, если кодирующую нуклеиновую кислоту конструируют путем простого связывания кодонов нуклеиновой кислоты, соответствующих каждой аминокислоте кодируемого целевого пептида, можно сконструировать несколько кодирующих нуклеиновых кислот для подлежащей кодированию одной целевой нуклеиновой кислоты. Поскольку одной стандартной аминокислоте соответствует в среднем от 1 до 6 кодонов нуклеиновых кислот, число возможных комбинаций кодонов нуклеиновых кислот увеличивается экспоненциально по мере увеличения длины аминокислотной последовательности. Однако не все эти комбинации одинаково важны. В общем, существует комбинация кодонов нуклеиновой кислоты, способная лучше экспрессировать целевой пептид, который подлежит кодированию в клетках, комбинация может варьироваться в зависимости от структуры более высокого порядка самой последовательности, типа целевой клетки, в которую необходимо ввести нуклеиновую кислоту и т.д. Обнаружение комбинации такого кодона нуклеиновой кислоты и указание обнаруженной комбинации в качестве последовательности кодирующей нуклеиновой кислоты называется оптимизацией по кодонам. Не обязательно существует только одна оптимизированная по кодонам последовательность для одного кодирующего целевого пептида, и может быть две или более последовательностей с оптимизированными кодонами.
[00407] В одном варианте осуществления кодирующая нуклеиновая кислота может иметь оптимизированную по кодонам последовательность ДНК и/или РНК. В другом варианте осуществления кодирующая нуклеиновая кислота может иметь не оптимизированные по кодонам последовательности ДНК и/или РНК.
[00408] оптимизация по кодонам кодирующей нуклеиновой кислоты 1 - рассмотрение структуры кодирующей нуклеиновой кислоты более высокого порядка
[00409] оптимизация по кодонам кодирующей нуклеиновой кислоты может быть выполнена с учетом структуры более высокого порядка самой последовательности нуклеиновой кислоты. В одном варианте осуществления кодон-оптимизированная нуклеиновая кислота может быть оптимизирована с учетом содержания GC в последовательности. В другом варианте осуществления последовательность кодирующей нуклеиновой кислоты может иметь содержание GC в диапазоне примерно менее 1%, примерно 1%, примерно 2%, примерно 3%, примерно 4%, примерно 5%, примерно 6%, примерно 7%, примерно 8%, примерно 9%, примерно 10%, примерно 11%, примерно 12%, примерно 13%, примерно 14%, примерно 15%, примерно 16%, примерно 17%, примерно 18%, примерно 19%, примерно 20%, примерно 21%, примерно 22%, примерно 23%, примерно 24%, примерно 25%, примерно 26%, примерно 27%, примерно 28%, примерно 29%, примерно 30%, примерно 31%, примерно 32%, примерно 33%, примерно 34%, примерно 35%, примерно 36%, примерно 37%, примерно 38%, примерно 39%, примерно 40%, примерно 41%, примерно 42%, примерно 43%, примерно 44%, примерно 45%, примерно 46%, примерно 47%, примерно 48%, примерно 49%, примерно 50%, примерно 51%, примерно 52%, примерно 53%, примерно 54%, примерно 55%, примерно 56%, примерно 57%, примерно 58%, примерно 59%, примерно 60%, примерно 61%, примерно 62%, примерно 63%, примерно 64%, примерно 65%, примерно 66%, примерно 67%, примерно 68%, примерно 69%, примерно 70%, примерно 71%, примерно 72%, примерно 73%, примерно 74%, примерно 75%, примерно 76%, примерно 77%, примерно 78%, примерно 79%, примерно 80%, примерно 81%, примерно 82%, примерно 83%, примерно 84%, примерно 85%, примерно 86%, примерно 87%, примерно 88%, примерно 89%, примерно 90%, примерно 91%, примерно 92%, примерно 93%, примерно 94%, примерно 95%, примерно 96%, примерно 97%, примерно 98%, примерно 99% или примерно 100%. Еще в одном варианте осуществления последовательность кодирующей нуклеиновой кислоты может иметь содержание GC в числовом диапазоне, выбранном в непосредственно предшествующем предложении. Например, последовательность кодирующей нуклеиновой кислоты может иметь содержание GC в диапазоне от примерно 20% до примерно 50%. Еще в одном варианте осуществления последовательность кодирующей нуклеиновой кислоты может иметь содержание GC меньше, чем значение, выбранное в непосредственно предшествующем предложении. Например, последовательность кодирующей нуклеиновой кислоты может иметь содержание GC менее примерно 25%.
[00410] оптимизация по кодонам кодирующей нуклеиновой кислоты 2 - рассмотрение целевых клеток для экспрессии
[00411] Оптимизация по кодонам кодирующей нуклеиновой кислоты может быть достигнута с учетом того, в какую клетку следует вводить и экспрессировать кодирующую нуклеиновую кислоту. В одном варианте осуществления оптимизация по кодонам кодирующей нуклеиновой кислоты может быть достигнута с учетом использования кодонов в прокариотических или эукариотических клетках. В другом варианте осуществления оптимизация по кодонам кодирующей нуклеиновой кислоты может быть достигнута с учетом использования кодонов клетками животных. Еще в одном варианте осуществления оптимизация по кодонам кодирующей нуклеиновой кислоты может быть достигнута с учетом использования кодонов млекопитающих. Еще в одном варианте осуществления оптимизация по кодонам кодирующей нуклеиновой кислоты может быть достигнута с учетом использования кодонов человека. Еще в одном варианте осуществления кодирующая нуклеиновая кислота может быть нуклеиновой кислотой, оптимизированной по кодонам E.coli. Еще в одном варианте осуществления кодирующая нуклеиновая кислота может представлять собой нуклеиновую кислоту с оптимизацией по кодонам млекопитающего. Еще в одном варианте осуществления кодирующая нуклеиновая кислота может быть кодон-оптимизированной нуклеиновой кислотой человека.
[00412] варианты кодирующих последовательностей нуклеиновых кислот
[00413] В одном варианте осуществления кодирующая нуклеиновая кислота может быть представлена последовательностью, выбранной из 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTCC GTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 248), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTCC GTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 249), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTCC GTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 250), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNGGATCGCATC ACCATCACCATCACGGATCCGATGATGATGACAAG-3' (SEQ ID NO: 251), 5'-ACGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNN NNAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNGGATCGCAT CACCATCACCATCACGGATCCGATGATGATGACAAG-3' (SEQ ID NO: 252), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNGGATCGCATC ACCATCACCATCACGGATCCGATGATGATGACAAG-3' (SEQ ID NO: 253), 5'-GGATCGCATCACCATCACCATCACGGATCCGATGATGATGACAAGNNNN NNAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNCGTAATGTT CCTCCTATCTTCAATGATGTTTATTGGATTGCATTC-3' (SEQ ID NO: 254), 5'-GGATCGCATCACCATCACCATCACGGATCCGATGATGATGACAAGNNNN NNAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNCGTAATGTT CCTCCTATCTTCAATGATGTTTATTGGATTGCATTC-3' (SEQ ID NO: 255), 5'-GGATCGCATCACCATCACCATCACGGATCCGATGATGATGACAAGNNNN NNAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNCGTAATGTT CCTCCTATCTTCAATGATGTTTATTGGATTGCATTC-3' (SEQ ID NO: 256), 5'-AAAACGACAAAGCAATCATTTGATTTAAGTGTAAAAGCTCAGTATNNNN NNAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTC CGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 257), 5'-AAAACGACAAAGCAATCATTTGATTTAAGTGTAAAAGCTCAGTATNNNN NNAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTC CGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 258), 5'-AAAACGACAAAGCAATCATTTGATTTAAGTGTAAAAGCTCAGTATNNNN NNAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTC CGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 259), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCTGCCGT TTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCCNNNNNNAAGNNN GTGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 260), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCTGCCGT TTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCCNNNNNNAAGTTCG TGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 261), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCTGCCGT TTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCCNNNNNNAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 262), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNCCT AAGTATGTGAAGCAGAATACACTGAAGCTGGCAACCNNNTGCCGTTTCCGT GGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 263), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 264), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 265), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 266), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGNN NGTGGCAGCTTGGACCCTGAAGGCAGCA-3' (SEQ ID NO: 267), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGTTC GTGGCAGCTTGGACCCTGAAGGCAGCA-3' (SEQ ID NO: 268), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGTAT GTGGCAGCTTGGACCCTGAAGGCAGCA-3' (SEQ ID NO: 269), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGNN NGTGGCAGCTTGGACCCTGAAGGCAGCACATCACCATCACCATCAC-3' (SEQ ID NO: 270), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGTTC GTGGCAGCTTGGACCCTGAAGGCAGCACATCACCATCACCATCAC-3' (SEQ ID NO: 271), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGTAT GTGGCAGCTTGGACCCTGAAGGCAGCACATCACCATCACCATCAC-3' (SEQ ID NO: 272), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGTTTCCGTGGAC TGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 273), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGTTTCCGTGGACT GATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 274), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGTTTCCGTGGACT GATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 275), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGT-3' (SEQ ID NO: 276), 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGT-3' (SEQ ID NO: 277), and 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNN NAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGT-3' (SEQ ID NO: 278).
[00414] В одном варианте осуществления кодирующая нуклеиновая кислота может быть представлена последовательностью РНК, эквивалентной последовательности, выбранной из SEQ ID NO: 248 - SEQ ID NO: 278.
[00415] В другом варианте осуществления кодирующая нуклеиновая кислота может представлять собой нуклеиновую кислоту, в которой по меньшей мере один кодон заменен кодоном, кодирующим ту же аминокислоту в последовательности, выбранной из SEQ ID NO: 248 - SEQ ID NO: 278. Например, кодирующая нуклеиновая кислота может представлять собой нуклеиновую кислоту, в которой первый кодон (нуклеиновые кислоты с первой по третью на 5'-конце) в последовательностях SEQ ID NO: 248 (т.е. CGT) заменен на CGC, CGG, CGA, AGA, или AGG.
[00416] Еще в одном варианте осуществления кодирующая нуклеиновая кислота может быть представлена последовательностью РНК, эквивалентной последовательности, выбранной из SEQ ID NO: 248 - SEQ ID NO: 278, в которой один или более кодонов заменены кодоном, кодирующим ту же самую аминокислоту.
[00417] фармацевтическая композиция, включающая нуклеиновую кислоту, кодирующую пептидную единицу и/или пептид
[00418] фармацевтическая композиция, включающая кодирующую нуклеиновую кислоту - обзор
[00419] Настоящее описание относится к фармацевтической композиции, включающей нуклеиновую кислоту, кодирующую пептидную единицу и/или пептид (т.е. кодирующую нуклеиновую кислоту). Для того чтобы доставить кодирующую нуклеиновую кислоту субъекту для проявления предполагаемого эффекта индукции иммунного ответа, необходимо приготовить кодирующую нуклеиновую кислоту соответствующим способом. Кодирующая нуклеиновая кислота может быть приготовлена известным способом, например, методами, описанными в WK KIM (2019, mRNA vaccine - new era in vaccinology, BRIC View 2019-R11), Zhang et al.(2019, Advances in mRNA Vaccines for Infectious Diseases, Frontiers in Immunology, Vol.10, Article 594), Reichmuth et al. (2016, mRNA vaccine delivery using lipid nanoparticles, Therapeutic Delivery, 7(5), 319-334), Miao et al. (2021, mRNA vaccine for cancer immunotherapy, Molecular Cancer, 20:41), Boen et al. (2021, Identification of T Cell Ligands in a Library of Peptides Covalently Attached to HLA-DR4, The Journal of Immunology, 165:2040-2047), Pardi et al.(2018, mRNA vaccines - a new era in vaccinology, Nature Reviews, Vol.17, 261-279) и в заявке на патент Кореи №10-2017-0054429, но способ ими не ограничивается.
[00420] Фармацевтическая композиция, включающая кодирующую нуклеиновую кислоту, может дополнительно включать адъюванты и/или дополнительные ингредиенты в дополнение к составу, кодирующему нуклеиновую кислоту. В одном варианте осуществления фармацевтическая композиция, включающая кодирующую нуклеиновую кислоту, включает следующее: готовые к применению кодирующие нуклеиновые кислоты; необязательно адъюванты; и, необязательно, дополнительные ингредиенты.
[00421] готовая к применению кодирующая нуклеиновая кислота
[00422] Готовая к применению кодирующая нуклеиновая кислота может быть приготовлена специалистами в данной области путем выбора подходящего средства доставки (вектора) для кодирующей нуклеиновой кислоты. Кодирующая нуклеиновая кислота может быть приготовлена с использованием вирусного вектора и/или невирусного вектора. В одном варианте осуществления готовая к применению кодирующая нуклеиновая кислота может включать вирусный вектор. В другом варианте осуществления готовая к применению кодирующая нуклеиновая кислота может включать невирусный вектор. В частности, невирусный вектор может включать липиды, полимеры и неорганические наночастицы, но не ограничивается ими.
[00423] Еще в одном варианте осуществления готовая к применению кодирующая нуклеиновая кислота может включать одно или более, выбранное из следующего:
[00424] депротеинизированную нуклеиновую кислоту; катионный пептид в комплексе с нуклеиновой кислотой (протамин); положительно заряженная катионная наноэмульсия масло-вода (катионная наноэмульсия); нуклеиновую кислоту, химически связанную с модифицированным дендримером и образующую комплекс с полиэтиленгликолем и ПЭГ-липидами (наночастица модифицированного дендримера); нуклеиновую кислоту в комплексе с протамином в ПЭГ-липидных наночастицах (протаминовая липосома); нуклеиновую кислоту в комплексе с катионным полимером (например, полиэтиленимином (PEI)) (катионный полимер); нуклеиновую кислоту в комплексе с катионными полимерами, такими как PEI и липидные компоненты (катионные полимерные липосомы); нуклеиновую кислоту в комплексе с полисахаридным полимером (например, хитозаном) (полисахаридные частицы); нуклеиновую кислоту в комплексе с полимерами катионных липидных наночастиц (катионные липидные наночастицы); нуклеиновую кислоту в комплексе с катионными липидами и холестерином (катионные липидно-холестериновые наночастицы); и нуклеиновую кислоту в комплексе с катионным липидом, холестерином и ПЭГ-липидом (наночастицы катионный липид-холестерин-ПЭГ).
[00425] Еще в одном варианте осуществления готовая к применению кодирующая нуклеиновая кислота может включать липидные наночастицы (LNP). В приведенном выше конкретном варианте осуществления липидные наночастицы могут представлять собой ионизируемые катионные липиды, фосфолипиды, холестерин и/или связанный с липидами полиэтиленгликоль. В частности, ионизируемый катионный липид может представлять собой одно или более из следующего: DLin-DMA; DLin-KC2-DMA; DLin-MC3-DMA; С12-200; сКК-Е12; производное DLin-MC3-DMA L319 (Alnylam and AlCana Technologies); C12-200 и производное cKK-E12 (Anderson Group); Липид вакцины против COVID-19 ALC-0315 и SM-102; ТТ3 и биоразлагаемое производное FTT5 (группа Dong); липиды витаминного происхождения ssPalmE и VcLNP; A9 (Acuitas); L5 (Moderna); липид А18; липид ATX (композиция LUNAR®; Arcturus); и LP01 (Intellia Therapeutics). В частности, фосфолипид может представлять собой одно или более из следующего: 1,2-диолеоил-sn-глицеро-3-фосфоэтаноламин (DOPE); и 1,2-дистеароил-sn-глицеро-3-фосфохолин (DSPC).
[00426] В конкретном варианте осуществления готовая к применению кодирующая нуклеиновая кислота может включать систему доставки на основе полимера. В конкретном варианте осуществления система доставки на основе полимера может включать один или более компонентов, выбранных из следующих: полиэтиленимин (PEI); полиамидоамин (PAMAM); полипропиленимин; и дендример на основе полимера.
[00427] Еще в одном варианте осуществления готовая к применению кодирующая нуклеиновая кислота может включать систему доставки на основе пептидов. В конкретном варианте осуществления система доставки на основе пептидов может включать протамин. В частности, готовая к применению кодирующая нуклеиновая кислота может представлять собой комплекс протамин-мРНК.
[00428] Еще в одном варианте осуществления готовая к применению кодирующая нуклеиновая кислота может включать катионные липидные составляющие липосомы, липоплексы и/или катионные эмульсии (CNE). В конкретном варианте осуществления катионный липид может представлять собой 1,2-ди-О-октадеценил-3-триметиламмонийпропан (ДОТМА) и/или 1,2-диолеоил-3-триметиламмонийпропан (DOTAP).
[00429] адьюванты
[00430] В одном варианте осуществления фармацевтическая композиция, включающая кодирующую нуклеиновую кислоту, может включать липидные наночастицы (LNP), соли алюминия, хлорид 1,2-диолеил-3-триметиламмонийпропан, адъювант MF59 (Novartis), CD70, лиганд CD40 (CD40L), TriMix, протамин, действующий через сигнальный путь TLR7, и/или монофосфориллипид A бактериального происхождения в качестве адъювантов.
[00431] ингредиенты
[00432] В одном варианте осуществления фармацевтическая композиция, включающая кодирующую нуклеиновую кислоту, может необязательно включать различные дополнительные ингредиенты. В другом варианте осуществления дополнительные ингредиенты могут представлять собой один или более компонентов, выбранных из следующего:
[00433] липиды; соли для балансировки кислотности организма; сахароза для сохранения стабильности при многократном замораживании-оттаивании; и вещества, повышающие стабильность вакцин
[00434] В частности, липид может представлять собой SM-102, PEG2000-DMG, DPSC, холестерин и/или ALC-0315, но не ограничивается ими. В частности, соль может представлять собой ацетат натрия, хлорид калия, дигидрофосфат калия, хлорид натрия и/или гидрофосфат натрия дигидрат, но не ограничивается ими. В частности, вещество, повышающее стабильность вакцины, может представлять собой уксусную кислоту, стабилизатор кислоты (трометамин) и/или этанол, но не ограничивается ими.
[00435] ПЕПТИД - СВОДНЫЕ ДАННЫЕ
[00436] Предложенный в настоящем описании пептид включает по меньшей мере одну пептидную единицу, и пептидная единица включает по меньшей мере один В-клеточный эпитоп и по меньшей мере один Th-эпитоп и может включать соответствующее количество вспомогательных частей. Пептидная единица представляет собой часть, предназначенную для равномерной индукции только заданного антитела, проявляя при этом определенный уровень иммуногенности в организме пациента. Кроме того, поскольку пептидная единица разработана с относительно небольшой длиной, она отличается простотой синтеза и низкой себестоимостью производства. Пептид обладает свойствами, подходящими для применения в качестве иммунотерапевтического средства, благодаря характеристикам пептидной единицы, описанным выше. В настоящем описании подробно раскрыты принципы конструирования пептида и пептидной единицы.
[00437] Названия каждой части описанного в настоящем документе пептида (например, вспомогательной части) даны для удобства пояснения. Соответственно, объем и название каждой части могут различаться в зависимости от точки зрения. Например, вспомогательная часть может называться защитной частью, фиктивной частью и/или линкером, но не ограничивается этим. В качестве другого примера, В-клеточный эпитоп может называться Th-эпитоп-защитным эпитопом, но не ограничиваясь этим.
[00438] Возможные примеры изобретения
[00439] Далее перечислены возможные примеры изобретения, предложенные в настоящем описании. Следующие Примеры, предложенные в этом разделе, просто соответствуют вариантам осуществления изобретения. Следовательно, изобретение, предложенное в настоящем описании, не может быть истолковано как ограниченное следующими примерами.
[00440] символы, используемые в каждом примере
[00441] Далее будут описаны символы, используемые для краткого описания каждого примера, в дополнение к номерам для различения каждого примера.
[00442] «В» обозначает В-клеточный эпитоп.«Т» обозначает Th-эпитоп.«А» обозначает вспомогательную часть (Auxiliary part). «U» обозначает пептидную единицу.
[00443] Когда каждый компонент связан знаком «-», этот компонент означает, что компоненты по обе стороны от «-» связаны напрямую или связаны через любой другой компонент.Например, когда он описывается как В-Т, он включает все пептиды, в которых В-клеточный эпитоп и Th-эпитоп связаны напрямую, и пептид, в котором В-клеточный эпитоп и Th-эпитоп связаны через любую другую последовательность.
[00444] При необходимости каждый компонент может быть помечен номером нижнего индекса, указывающим, что эти два компонента различны. Например, при выражении как B1-B2-T, B1 и B2 представлены разные B-клеточные эпитопы.
[00445] Приведенные выше символы предназначены только для схематического описания примеров, и их не следует интерпретировать, ограничивая примеры этими символами.
[00446] пептидная единица 1
[00447] Пример 1. Пептидная единица, которая может индуцировать гуморальный иммунитет
[00448] Пептидная единица, которая может индуцировать гуморальный иммунитет за счет распознавания CD4+Т-клетками, включающая следующее: по меньшей мере один Th-эпитоп; и по меньшей мере один В-клеточный эпитоп, в котором пептидная единица отличается тем, что длина Th-эпитопа соответствует 8-меру, 9-меру, 10-меру, 11-меру, 12-меру, 13-меру, 14-меру, 15-меру, 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру или 30-меру; длина В-клеточного эпитопа соответствует 8-меру, 9-меру, 10-меру, 11-меру, 12-меру, 13-меру, 14-меру, 15-меру, 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру или 32-меру; и длина пептидной единицы соответствует 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру, 32-меру, 33-меру, 34-меру, 35-меру, 36-меру, 37-меру, 38-меру, 39-меру, 40-меру, 41-меру, 42-меру, 43-меру, 44-меру, 45-меру, 46-меру, 47-меру, 48-меру, 49-меру, 50-меру, 51-меру, 52-меру, 53-меру, 54-меру, 55-меру, 56-меру, 57-меру, 58-меру, 59-меру, 60-меру, 61-меру, 62-меру, 63-меру, 64-меру, 65-меру, 66-меру, 67-меру, 68-меру, 69-меру, 70-меру, 71-меру, 72-меру, 73-меру, 74-меру, 75-меру, 76-меру, 77-меру, 78-меру, 79-меру, 80-меру, 81-меру, 82-меру, 83-меру, 84-меру, 85-меру, 86-меру, 87-меру, 88-меру, 89-меру, 90-меру, 91-меру, 92-меру, 93-меру, 94-меру, 95-меру, 96-меру, 97-меру, 98-меру, 99-меру или 100-меру.
[00449] Пример 2. Пептидная единица, индуцирующая образование антител, нацеленных на аполипопротеин B-100.
[00450] Пептидная единица, которая может индуцировать гуморальный иммунитет за счет распознавания CD4+Т-клетками, включающая следующее: по меньшей мере один Th-эпитоп; и по меньшей мере один В-клеточный эпитоп, в котором пептидная единица отличается тем, что длина Th-эпитопа соответствует 8-меру, 9-меру, 10-меру, 11-меру, 12-меру, 13-меру, 14-меру, 15-меру, 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру или 30-меру; В-клеточный эпитоп способен индуцировать антитела, нацеленные на аполипопротеин В-100; и длина пептидной единицы соответствует 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру, 32-меру, 33-меру, 34-меру, 35-меру, 36-меру, 37-меру, 38-меру, 39-меру, 40-меру, 41-меру, 42-меру, 43-меру, 44-меру, 45-меру, 46-меру, 47-меру, 48-меру, 49-меру, 50-меру, 51-меру, 52-меру, 53-меру, 54-меру, 55-меру, 56-меру, 57-меру, 58-меру, 59-меру, 60-меру, 61-меру, 62-меру, 63-меру, 64-меру, 65-меру, 66-меру, 67-меру, 68-меру, 69-меру, 70-меру, 71-меру, 72-меру, 73-меру, 74-меру, 75-меру, 76-меру, 77-меру, 78-меру, 79-меру, 80-меру, 81-меру, 82-меру, 83-меру, 84-меру, 85-меру, 86-меру, 87-меру, 88-меру, 89-меру, 90-меру, 91-меру, 92-меру, 93-меру, 94-меру, 95-меру, 96-меру, 97-меру, 98-меру, 99-меру или 100-меру.
[00451] Пример 3. Пептидная единица, индуцирующая антитело, которое нацелено на аполипопротеин B-100, включенный в LDL и/или VLDL
[00452] Пептидная единица примера 2, где В-клеточный эпитоп отличается тем, что он индуцирует антитело, нацеленное на сайт, выбранный из следующего: открытый сайт аполипопротеина В-100, включенный в липопротеин низкой плотности (LDL); и открытый сайт аполипопротеина B-100, включенный в липопротеин сверхнизкой плотности (VLDL).
[00453] Пример 4. Ограничение по типу B
[00454] Пептидная единица примера 2, где В-клеточный эпитоп представляет собой фрагмент аполипопротеина В-100 и/или мимотоп аполипопротеина В-100.
[00455] Пример 5. Ограничение по последовательности B
[00456] Пептидная единица примера 4, где В-клеточный эпитоп представляет собой последовательность, выбранную из группы, состоящей из RNVPPIFNDVYWIAF (SEQ ID NO: 6), CRFRGLISLSQVYLS (SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8), RFRGLISLSQVYLDP (SEQ ID NO: 221), и SVCGCPVGHHDVVGL (SEQ ID NO: 222), или эпитоп, включенный в последовательность, выбранную из SEQ ID NO: 6-8 и 221-222.
[00457] Пример 6. Ограничение по длине полноразмерной последовательности пептидной единицы
[00458] Пептидная единица любого из примеров 1-5, где пептидная единица отличается тем, что длина пептидной единицы составляет от 23-мера до 71-мера или от 26-мера до 50-мера.
[00459] пептидная единица 2 - единица А
[00460] Пример 7, B-T и T-B
[00461] Пептидная единица любого из примеров 1-4, где пептидная единица включает один В-клеточный эпитоп и один Th-эпитоп.
[00462] Пример 8. Ограничение по последовательности B, ограничение по длине T и ограничение по длине U
[00463] Пептидная единица примера 7, где длина пептидной единицы составляет от 26-мера до 45-мера; В-клеточный эпитоп выбран из RNVPPIFNDVYWIAF (SEQ ID NO: 6), CRFRGLISLSQVYLS (SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8), RFRGLISLSQVYLDP (SEQ ID NO: 221), и SVCGCPVGHHDVVGL (SEQ ID NO: 222); и длина Th-эпитопа составляет от 11-мера до 13-мера.
[00464] Пример 9. Ограничение по последовательности T
[00465] Пептидная единица любого из примеров 7 и 8, где Th-эпитоп отличается тем, что он выбран из группы, состоящей из следующего: K(Cha)K, (Cha)VAAWTLKAA (SEQ ID NO: 1); PKYVKQNTLKLAT (SEQ ID NO: 2); ILMQYIKANSKFIGI (SEQ ID NO: 3); QSIALSSLMVAQAIP (SEQ ID NO: 4); ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 5); PLGFFPDHQL (SEQ ID NO: 162); WPEANQVGAGAFGPGF (SEQ ID NO: 163); MQWNSTALHQALQDP (SEQ ID NO: 164); MQWNSTTFHQTLQDPRVRGLYFPAGG (SEQ ID NO: 165); FFLLTRILTI (SEQ ID NO: 166); FFLLTRILTIPQSLD (SEQ ID NO: 167); TSLNFLGGTTVCLGQ (SEQ ID NO: 168); QSPTSNHSPTSCPPIC (SEQ ID NO: 169); IIFLFILLLCLIFLLVLLD (SEQ ID NO: 170); CTTPAQGNSMFPSC (SEQ ID NO: 171); CTKPTDGN (SEQ ID NO: 172); WASVRFSW (SEQ ID NO: 173); LLPIFFCLW (SEQ ID NO: 174); MDIDPYKEFGATVELLSFLP (SEQ ID NO: 175); FLPSDFFPSV (SEQ ID NO: 176); RDLLDTASALYREALESPEH (SEQ ID NO: 177); PHHTALRQAILCWGELMTLA (SEQ ID NO: 178); GRETVIEYLVSFGVW (SEQ ID NO: 179); EYLVSFGVWIRTPPA (SEQ ID NO: 180); VSFGVWIRTPPAYRPPNAPI (SEQ ID NO: 181); TVVRRRGRSP (SEQ ID NO: 182); VGPLTVNEKRRLKLI (SEQ ID NO: 183); RHYLHTLWKAGILYK (SEQ ID NO: 184); ESRLVVDFSQFSRGN (SEQ ID NO: 185); LQSLTNLLSSNLSWL (SEQ ID NO: 186); SSNLSWLSLDVSAAF (SEQ ID NO: 187); LHLYSHPIILGFRKI (SEQ ID NO: 188); KQCFRKLPVNRPIDW (SEQ ID NO: 189); LCQVFADATPTGWGL (SEQ ID NO: 190); AANWILRGTSFVYVP (SEQ ID NO: 191); и EIRLKVFVLGGCRHK (SEQ ID NO: 192), в которых (Cha) обозначает L-циклогексилаланины.
[00466] Пример 10, BAT и TAB
[00467] Пептидная единица любого из примеров 7 и 8, где эта пептидная единица отличается тем, что она дополнительно включает вспомогательную часть, и что вспомогательная часть связана между В-клеточным эпитопом и Th-эпитопом.
[00468] Пример 11. Ограничение по А, включающей нестандартную аминокислоту.
[00469] Пептидная единица Примера 10, где вспомогательная часть включает одну или более нестандартных аминокислот.
[00470] Пример 12. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00471] Пептидная единица Примера 11, где вспомогательная часть выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00472] Пример 13. Ограничение по последовательности А, не включающей нестандартную аминокислоту.
[00473] Пептидная единица Примера 10, где вспомогательная часть выбрана из CR, HHHHHH (SEQ ID NO: 53) и RRRRRR (SEQ ID NO: 159).
[00474] пептидная единица 3 - единица D
[00475] Пример 14, B-T1-T2 и T2-T1-B
[00476] Пептидная единица любого из примеров 1-4, где пептидная единица отличается тем, что она включает один В-клеточный эпитоп и два Th-эпитопа, которые обозначаются как первый Th-эпитоп и второй Th-эптоп, соответственно, и тем, что первый Th-эпитоп связан между В-клеточным эпитопом и вторым Th-эпитопом.
[00477] Пример 15. Ограничение по последовательности B, ограничение по длине T и ограничение по длине U
[00478] Пептидная единица Примера 14, где длина пептидной единицы составляет от 37-мера до 50-мера; В-клеточный эпитоп выбран из RNVPPIFNDVYWIAF (SEQ ID NO: 6), CRFRGLISLSQVYLS (SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8), RFRGLISLSQVYLDP (SEQ ID NO: 221), и SVCGCPVGHHDVVGL (SEQ ID NO: 222); и длина первого Th-эпитопа и второго Th-эпитопа составляет от 11-мера до 13-мера, соответственно.
[00479] Пример 16. Ограничение по последовательности T
[00480] Пептидная единица любого из примеров 14 и 15, где Th-эпитоп отличается тем, что он выбран из группы, состоящей из следующего: K(Cha)VAAWTLKAA (SEQ ID NO: 1); PKYVKQNTLKLAT (SEQ ID NO: 2); ILMQYIKANSKFIGI (SEQ ID NO: 3); QSIALSSLMVAQAIP (SEQ ID NO: 4); ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 5); PLGFFPDHQL (SEQ ID NO: 162); WPEANQVGAGAFGPGF (SEQ ID NO: 163); MQWNSTALHQALQDP (SEQ ID NO: 164); MQWNSTTFHQTLQDPRVRGLYFPAGG (SEQ ID NO: 165); FFLLTRILTI (SEQ ID NO: 166); FFLLTRILTIPQSLD (SEQ ID NO: 167); TSLNFLGGTTVCLGQ (SEQ ID NO: 168); QSPTSNHSPTSCPPIC (SEQ ID NO: 169); IIFLFILLLCLIFLLVLLD (SEQ ID NO: 170); CTTPAQGNSMFPSC (SEQ ID NO: 171); CTKPTDGN (SEQ ID NO: 172); WASVRFSW (SEQ ID NO: 173); LLPIFFCLW (SEQ ID NO: 174); MDIDPYKEFGATVELLSFLP (SEQ ID NO: 175); FLPSDFFPSV (SEQ ID NO: 176); RDLLDTASALYREALESPEH (SEQ ID NO: 177); PHHTALRQAILCWGELMTLA (SEQ ID NO: 178); GRETVIEYLVSFGVW (SEQ ID NO: 179); EYLVSFGVWIRTPPA (SEQ ID NO: 180); VSFGVWIRTPPAYRPPNAPI (SEQ ID NO: 181); TVVRRRGRSP (SEQ ID NO: 182); VGPLTVNEKRRLKLI (SEQ ID NO: 183); RHYLHTLWKAGILYK (SEQ ID NO: 184); ESRLVVDFSQFSRGN (SEQ ID NO: 185); LQSLTNLLSSNLSWL (SEQ ID NO: 186); SSNLSWLSLDVSAAF (SEQ ID NO: 187); LHLYSHPIILGFRKI (SEQ ID NO: 188); KQCFRKLPVNRPIDW (SEQ ID NO: 189); LCQVFADATPTGWGL (SEQ ID NO: 190); AANWILRGTSFVYVP (SEQ ID NO: 191); и EIRLKVFVLGGCRHK (SEQ ID NO: 192), где а обозначает D-форму аланина, (Cha) обозначает L-циклогексилаланины и Z обозначает 6-аминогексановую кислоту.
[00481] Пример 17, BAT1-T2 и T2-T1AB
[00482] Пептидная единица любого из примеров 14 и 15, где пептидная единица отличается тем, что она дополнительно включает вспомогательную часть, и вспомогательная часть связана между В-клеточным эпитопом и первым Th-эпитопом.
[00483] Пример 18, B-T1AT2 и T2AT1-B
[00484] Пептидная единица любого из примеров 14 и 15, где пептидная единица отличается тем, что она дополнительно включает вспомогательную часть, и вспомогательная часть связана между первым Th-эпитопом и вторым Th-эпитопом.
[00485] Пример 19. Ограничение по А, включающей нестандартную аминокислоту.
[00486] Пептидная единица любого из примеров 17 и 18, где пептидная единица отличается тем, что вспомогательная часть включает одну или более нестандартных аминокислот.
[00487] Пример 20. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00488] Пептидная единица примера 19, где вспомогательная часть выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00489] Пример 21. Ограничение по последовательности А, не включающей нестандартную аминокислоту.
[00490] Пептидная единица любого из примеров 17 и 18, где пептидная единица отличается тем, что вспомогательная часть выбрана из CR, HHHHHH (SEQ ID NO: 53), и RRRRRR (SEQ ID NO: 159).
[00491] Пример 22, BAT1AT2 и T2AT1AB
[00492] Пептидная единица любого из примеров 14 и 15, где пептидная единица отличается тем, что она дополнительно включает первую вспомогательную часть и вторую вспомогательную часть, и что первая вспомогательная часть связана между В-клеточным эпитопом и Th эпитоп, а вторая вспомогательная часть связана между первым Th эпитопом и вторым Th эпитопом.
[00493] Пример 23. Ограничение по А, включающей нестандартную аминокислоту.
[00494] Пептидная единица из Примера 22, где первая вспомогательная часть и/или вторая вспомогательная часть включают одну или более нестандартных аминокислот.
[00495] Пример 24. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00496] Пептидная единица примера 23, где вспомогательная часть, включающая одну или более нестандартных аминокислот, независимо выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00497] Пример 25. Ограничение по последовательности А, не включающей нестандартную аминокислоту.
[00498] Пептидная единица Примера 22, где первая вспомогательная часть и/или вторая вспомогательная часть независимо выбраны из CR, HHHHHH (SEQ ID NO: 53) и RRRRRR (SEQ ID NO: 159).
[00499] пептидная единица 4 - единица B
[00500] Пример 26, B1-B2-T и T-B2-B1
[00501] Пептидная единица любого из примеров 1-4, где пептидная единица отличается тем, что включает два В-клеточных эпитопа, которые называются первым В-клеточным эпитопом и вторым В-клеточным эпитопом, соответственно, и что второй В-клеточный эпитоп связан между первым В-клеточным эпитопом и Th-эпитопом.
[00502] Пример 27. Ограничение по последовательности B, ограничение по длине T и ограничение по длине U
[00503] Пептидная единица примера 26, где длина пептидной единицы составляет от 45-мера до 50-мера; первый В-клеточный эпитоп и второй В-клеточный эпитоп независимо выбраны из RNVPPIFNDVYWIAF (SEQ ID NO: 6), CRFRGLISLSQVYLS (SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8), RFRGLISLSQVYLDP (SEQ ID NO: 221), and SVCGCPVGHHDVVGL (SEQ ID NO: 222); и длина Th-эпитопа составляет от 11-мера до 13-мера.
[00504] Пример 28. Ограничение по последовательности T
[00505] Пептидная единица любого из примеров 26 и 27, где Th-эпитоп отличается тем, что он выбран из группы, состоящей из следующего: K(Cha)VAAWTLKAA (SEQ ID NO: 1); PKYVKQNTLKLAT (SEQ ID NO: 2); ILMQYIKANSKFIGI (SEQ ID NO: 3); QSIALSSLMVAQAIP (SEQ ID NO: 4); ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 5); PLGFFPDHQL (SEQ ID NO: 162); WPEANQVGAGAFGPGF (SEQ ID NO: 163); MQWNSTALHQALQDP (SEQ ID NO: 164); MQWNSTTFHQTLQDPRVRGLYFPAGG (SEQ ID NO: 165); FFLLTRILTI (SEQ ID NO: 166); FFLLTRILTIPQSLD (SEQ ID NO: 167); TSLNFLGGTTVCLGQ (SEQ ID NO: 168); QSPTSNHSPTSCPPIC (SEQ ID NO: 169); IIFLFILLLCLIFLLVLLD (SEQ ID NO: 170); CTTPAQGNSMFPSC (SEQ ID NO: 171); CTKPTDGN (SEQ ID NO: 172); WASVRFSW (SEQ ID NO: 173); LLPIFFCLW (SEQ ID NO: 174); MDIDPYKEFGATVELLSFLP (SEQ ID NO: 175); FLPSDFFPSV (SEQ ID NO: 176); RDLLDTASALYREALESPEH (SEQ ID NO: 177); PHHTALRQAILCWGELMTLA (SEQ ID NO: 178); GRETVIEYLVSFGVW (SEQ ID NO: 179); EYLVSFGVWIRTPPA (SEQ ID NO: 180); VSFGVWIRTPPAYRPPNAPI (SEQ ID NO: 181); TVVRRRGRSP (SEQ ID NO: 182); VGPLTVNEKRRLKLI (SEQ ID NO: 183); RHYLHTLWKAGILYK (SEQ ID NO: 184); ESRLVVDFSQFSRGN (SEQ ID NO: 185); LQSLTNLLSSNLSWL (SEQ ID NO: 186); SSNLSWLSLDVSAAF (SEQ ID NO: 187); LHLYSHPIILGFRKI (SEQ ID NO: 188); KQCFRKLPVNRPIDW (SEQ ID NO: 189); LCQVFADATPTGWGL (SEQ ID NO: 190); AANWILRGTSFVYVP (SEQ ID NO: 191); и EIRLKVFVLGGCRHK (SEQ ID NO: 192), где а обозначает D-форму аланина, (Cha) обозначает L-циклогексилаланины и Z обозначает 6-аминогексановую кислоту.
[00506] Пример 29, B1-B2AT и TAB2-B1
[00507] Пептидная единица любого из примеров 26 и 27, где пептидная единица отличается тем, что она дополнительно включает вспомогательную часть, и вспомогательная часть связана между вторым В-клеточным эпитопом и Th-эпитопом.
[00508] Пример 30, B1AB2-T и T-B2AB1
[00509] Пептидная единица любого из примеров 26 и 27, где пептидная единица отличается тем, что она дополнительно включает вспомогательную часть, и вспомогательная часть связана между первым В-клеточным эпитопом и вторым В-клеточным эпитопом.
[00510] Пример 31. Ограничение по А, включающей нестандартную аминокислоту.
[00511] Пептидная единица любого из примеров 29 и 30, где пептидная единица отличается тем, что вспомогательная часть включает одну или более нестандартных аминокислот.
[00512] Пример 32. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00513] Пептидная единица примера 31, где вспомогательная часть выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00514] Пример 33, А, Ограничение по последовательности, не включающей нестандартную аминокислоту.
[00515] Пептидная единица любого из примеров 29 и 30, где пептидная единица отличается тем, что вспомогательная часть выбрана из CR, HHHHHH (SEQ ID NO: 53) и RRRRRR (SEQ ID NO: 159).
[00516] Пример 34, B1A1B2A2T и TA2B2A1B1.
[00517] Пептидная единица любого из примеров 26 и 27, где пептидная единица отличается тем, что она дополнительно включает первую вспомогательную часть и вторую вспомогательную часть, и первая вспомогательная часть связана между первым В-клеточным эпитопом и вторым В-клеточный эпитоп, а вторая вспомогательная часть связана между вторым В-клеточным эпитопом и Th-эпитопом.
[00518] Пример 35. Ограничение по А, включающей нестандартную аминокислоту.
[00519] Пептидная единица Примера 34, где первая вспомогательная часть и/или вторая вспомогательная часть включают одну или более нестандартных аминокислот.
[00520] Пример 36. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00521] Пептидная единица Примера 35, где пептидная единица отличается тем, что вспомогательные части, включающие одну или более нестандартных аминокислот, каждая независимо выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00522] Пример 37. Ограничение по последовательности А, не включающей нестандартную аминокислоту.
[00523] Пептидная единица Примера 34, где пептидная единица отличается тем, что первая вспомогательная часть и/или вторая вспомогательная часть, каждая, независимо выбраны из CR, HHHHHH (SEQ ID NO: 53) и RRRRRR (SEQ ID NO: 159).
[00524] пептидная единица 5 - единица С
[00525] Пример 38, В1-Т-В2
[00526] Пептидная единица любого из примеров 1-4, где пептидная единица отличается тем, что она включает два В-клеточных эпитопа, которые называются первым В-клеточным эпитопом и вторым В-клеточным эпитопом, и один Th-эпитоп, и Th-эпитоп связан между первым В-клеточным эпитопом и вторым В-клеточным эпитопом.
[00527] Пример 39. Ограничение по последовательности B, ограничение по длине T и ограничение по длине U
[00528] Пептидная единица Примера 38, где пептидная единица отличается тем, что длина пептидной единицы составляет от 45-мера до 50-мера; каждый из первого В-клеточного эпитопа и второго В-клеточного эпитопа независимо выбран из RNVPPIFNDVYWIAF (SEQ ID NO: 6), CRFRGLISLSQVYLS (SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8), RFRGLISLSQVYLDP (SEQ ID NO: 221), и SVCGCPVGHHDVVGL (SEQ ID NO: 222); и длина Th-эпитопа составляет от 11-мера до 13-мера.
[00529] Пример 40. Ограничение по последовательности T
[00530] Пептидная единица любого из примеров 38 и 39, где Th-эпитоп отличается тем, что он выбран из группы, состоящей из следующего: K(Cha)VAAWTLKAA (SEQ ID NO: 1); PKYVKQNTLKLAT (SEQ ID NO: 2); ILMQYIKANSKFIGI (SEQ ID NO: 3); QSIALSSLMVAQAIP (SEQ ID NO: 4); ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 5); PLGFFPDHQL (SEQ ID NO: 162); WPEANQVGAGAFGPGF (SEQ ID NO: 163); MQWNSTALHQALQDP (SEQ ID NO: 164); MQWNSTTFHQTLQDPRVRGLYFPAGG (SEQ ID NO: 165); FFLLTRILTI (SEQ ID NO: 166); FFLLTRILTIPQSLD (SEQ ID NO: 167); TSLNFLGGTTVCLGQ (SEQ ID NO: 168); QSPTSNHSPTSCPPIC (SEQ ID NO: 169); IIFLFILLLCLIFLLVLLD (SEQ ID NO: 170); CTTPAQGNSMFPSC (SEQ ID NO: 171); CTKPTDGN (SEQ ID NO: 172); WASVRFSW (SEQ ID NO: 173); LLPIFFCLW (SEQ ID NO: 174); MDIDPYKEFGATVELLSFLP (SEQ ID NO: 175); FLPSDFFPSV (SEQ ID NO: 176); RDLLDTASALYREALESPEH (SEQ ID NO: 177); PHHTALRQAILCWGELMTLA (SEQ ID NO: 178); GRETVIEYLVSFGVW (SEQ ID NO: 179); EYLVSFGVWIRTPPA (SEQ ID NO: 180); VSFGVWIRTPPAYRPPNAPI (SEQ ID NO: 181); TVVRRRGRSP (SEQ ID NO: 182); VGPLTVNEKRRLKLI (SEQ ID NO: 183); RHYLHTLWKAGILYK (SEQ ID NO: 184); ESRLVVDFSQFSRGN (SEQ ID NO: 185); LQSLTNLLSSNLSWL (SEQ ID NO: 186); SSNLSWLSLDVSAAF (SEQ ID NO: 187); LHLYSHPIILGFRKI (SEQ ID NO: 188); KQCFRKLPVNRPIDW (SEQ ID NO: 189); LCQVFADATPTGWGL (SEQ ID NO: 190); AANWILRGTSFVYVP (SEQ ID NO: 191); и EIRLKVFVLGGCRHK (SEQ ID NO: 192), где а обозначает D-форму аланина, (Cha) обозначает L-циклогексилаланины и Z обозначает 6-аминогексановую кислоту.
[00531] Пример 41, B1AT-B2 и B2-TAB1.
[00532] Пептидная единица любого из примеров 38 и 39, где пептидная единица отличается тем, что она дополнительно включает вспомогательную часть, и вспомогательная часть связана между первым В-клеточным эпитопом и Th-эпитопом.
[00533] Пример 42. Ограничение по А, включающей нестандартную аминокислоту.
[00534] Пептидная единица Примера 41, где пептидная единица отличается тем, что вспомогательная часть включает одну или более нестандартных аминокислот,
[00535] Пример 43. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00536] Пептидная единица примера 42, где пептидная единица отличается тем, что вспомогательная часть выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00537] Пример 44. Ограничение по последовательности А, не включающей нестандартную аминокислоту.
[00538] Пептидная единица примера 41, где пептидная единица отличается тем, что вспомогательная часть выбрана из CR, HHHHHH (SEQ ID NO: 53), и RRRRRR (SEQ ID NO: 159).
[00539] Пример 45, B1A1TA2B2
[00540] Пептидная единица любого из примеров 38 и 39, где пептидная единица отличается тем, что она дополнительно включает первую вспомогательную часть и вторую вспомогательную часть; первая вспомогательная часть связана между первым В-клеточным эпитопом и Th-эпитопом; и вторая вспомогательная часть связана между вторым В-клеточным эпитопом и Th-эпитопом.
[00541] Пример 46. Ограничение по А, включающей нестандартную аминокислоту.
[00542] Пептидная единица из Примера 45, где пептидная единица отличается тем, что первая вспомогательная часть и/или вторая вспомогательная часть включают одну или более нестандартных аминокислот.
[00543] Пример 47. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00544] Пептидная единица Примера 46, где пептидная единица отличается тем, что вспомогательная часть, включающая одну или более нестандартных аминокислот, каждая независимо выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00545] Пример 48. Ограничение по последовательности А, не включающей нестандартную аминокислоту.
[00546] Пептидная единица Примера 45, где пептидная единица отличается тем, что первая вспомогательная часть и/или вторая вспомогательная часть, каждая независимо, выбрана из CR, HHHHHH (SEQ ID NO: 53), и RRRRRR (SEQ ID NO: 159).
[00547] пептидная единица 6 - единица E
[00548] Пример 49, В1-Т1-Т2-В2
[00549] Пептидная единица любого из примеров 1-4, где пептидная единица отличается тем, что она включает два В-клеточных эпитопа, каждый из которых обозначается как первый В-клеточный эпитоп и второй В-клеточный эпитоп, и два Th-эпитопа, каждый из которых обозначается как первый Th-эпитоп и второй Th-эпитоп, первый В-клеточный эпитоп, первый Th-эпитоп, второй Th-эпитоп и второй В-клеточный эпитоп, последовательно связаны в направлении от N-конца к С-концу.
[00550] Пример 50, ограничение по последовательности B, ограничение по длине T и ограничение по длине U
[00551] Пептидная единица примера 49, где пептидная единица отличается тем, что длина пептидной единицы составляет от 52-мера до 90-мера; каждый из первого В-клеточного эпитопа и второго В-клеточного эпитопа независимо выбран из RNVPPIFNDVYWIAF (SEQ ID NO: 6), CRFRGLISLSQVYLS (SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8), RFRGLISLSQVYLDP (SEQ ID NO: 221), и SVCGCPVGHHDVVGL (SEQ ID NO: 222); и длина каждого из первого Th-эпитопа и второго Th-эпитопа составляет от 11-мера до 13-мера.
[00552] Пример 51. Ограничение по последовательности T
[00553] Пептидная единица примера 50, где первый Th-эпитоп и второй Th-эпитоп отличается тем, что каждый из них независимо выбран из группы, состоящей из следующего: K(Cha)VAAWTLKAA (SEQ ID NO: 1); PKYVKQNTLKLAT (SEQ ID NO: 2); ILMQYIKANSKFIGI (SEQ ID NO: 3); QSIALSSLMVAQAIP (SEQ ID NO: 4); ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 5); PLGFFPDHQL (SEQ ID NO: 162); WPEANQVGAGAFGPGF (SEQ ID NO: 163); MQWNSTALHQALQDP (SEQ ID NO: 164); MQWNSTTFHQTLQDPRVRGLYFPAGG (SEQ ID NO: 165); FFLLTRILTI (SEQ ID NO: 166); FFLLTRILTIPQSLD (SEQ ID NO: 167); TSLNFLGGTTVCLGQ (SEQ ID NO: 168); QSPTSNHSPTSCPPIC (SEQ ID NO: 169); IIFLFILLLCLIFLLVLLD (SEQ ID NO: 170); CTTPAQGNSMFPSC (SEQ ID NO: 171); CTKPTDGN (SEQ ID NO: 172); WASVRFSW (SEQ ID NO: 173); LLPIFFCLW (SEQ ID NO: 174); MDIDPYKEFGATVELLSFLP (SEQ ID NO: 175); FLPSDFFPSV (SEQ ID NO: 176); RDLLDTASALYREALESPEH (SEQ ID NO: 177); PHHTALRQAILCWGELMTLA (SEQ ID NO: 178); GRETVIEYLVSFGVW (SEQ ID NO: 179); EYLVSFGVWIRTPPA (SEQ ID NO: 180); VSFGVWIRTPPAYRPPNAPI (SEQ ID NO: 181); TVVRRRGRSP (SEQ ID NO: 182); VGPLTVNEKRRLKLI (SEQ ID NO: 183); RHYLHTLWKAGILYK (SEQ ID NO: 184); ESRLVVDFSQFSRGN (SEQ ID NO: 185); LQSLTNLLSSNLSWL (SEQ ID NO: 186); SSNLSWLSLDVSAAF (SEQ ID NO: 187); LHLYSHPIILGFRKI (SEQ ID NO: 188); KQCFRKLPVNRPIDW (SEQ ID NO: 189); LCQVFADATPTGWGL (SEQ ID NO: 190); AANWILRGTSFVYVP (SEQ ID NO: 191); и EIRLKVFVLGGCRHK (SEQ ID NO: 192), где а обозначает D-форму аланина, (Cha) обозначает L-циклогексилаланины и Z обозначает 6-аминогексановую кислоту.
[00554] Пример 52, B1AT1-T2-B2 и B2-T2-T1AB1.
[00555] Пептидная единица любого из примеров 49 и 50, где пептидная единица отличается тем, что она дополнительно включает вспомогательную часть, и вспомогательная часть связана между первым В-клеточным эпитопом и первым Th-эпитопом.
[00556] Пример 53, B1-T1AT2-B2
[00557] Пептидная единица любого из примеров 49 и 50, где пептидная единица отличается тем, что она дополнительно включает вспомогательную часть, и вспомогательная часть связана между первым Th-эпитопом и вторым Th-эпитопом.
[00558] Пример 54, B1-T1-T2AB2 и B2AT2-T1-B1.
[00559] Пептидная единица любого из примеров 49 и 50, где пептидная единица отличается тем, что она дополнительно включает вспомогательную часть, и вспомогательная часть связана между вторым Th-эпитопом и вторым В-клеточным эпитопом.
[00560] Пример 55, Ограничение по А, включающей нестандартную аминокислоту.
[00561] Пептидная единица любого из примеров 52-54, где пептидная единица отличается тем, что вспомогательная часть включает одну или более нестандартных аминокислот.
[00562] Пример 56. Ограничение по А, включающей нестандартную аминокислоту.
[00563] Пептидная единица любого из примеров 52-54, где пептидная единица отличается тем, что вспомогательная часть выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00564] Пример 57. Ограничение последовательности А, не включающей нестандартную аминокислоту.
[00565] Пептидная единица любого из примеров 52-54, где пептидная единица отличается тем, что вспомогательная часть выбрана из CR, HHHHHH (SEQ ID NO: 53), и RRRRRR (SEQ ID NO: 159).
[00566] Пример 58, B1A1T1A2T2-B2 и B2-T2A2T1A1B1.
[00567] Пептидная единица любого из примеров 49 и 50, где пептидная единица отличается тем, что она дополнительно включает первую вспомогательную часть и вторую вспомогательную часть, и вспомогательная часть связана между первым В-клеточным эпитопом и первым Th эпитопом, а вторая вспомогательная часть связана между первым Th-эпитопом и вторым Th-эпитопом.
[00568] Пример 59, B1A1T1-T2A2B2
[00569] Пептидная единица любого из примеров 49 и 50, где пептидная единица отличается тем, что она дополнительно включает первую вспомогательную часть и вторую вспомогательную часть, и вспомогательная часть связана между первым В-клеточным эпитопом и первым Th эпитопом, а вторая вспомогательная часть связана между вторым Th-эпитопом и вторым В-клеточным эпитопом.
[00570] Пример 60, B1-T1A1T2A2B2 и B2-T2A2T1A1B1.
[00571] Пептидная единица любого из примеров 49 и 50, где пептидная единица отличается тем, что она дополнительно включает первую вспомогательную часть и вторую вспомогательную часть, и вспомогательная часть связана между первым Th-эпитопом и вторым Th-эпитопом, первый Th-эпитоп и второй Th-эпитоп связаны между вторым Th-эпитопом и вторым В-клеточным эпитопом.
[00572] Пример 61. Ограничение по А, включающей нестандартную аминокислоту.
[00573] Пептидная единица любого из примеров 58-60, где пептидная единица отличается тем, что первая вспомогательная часть и/или вторая вспомогательная часть включают одну или более нестандартных аминокислот.
[00574] Пример 62. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00575] Пептидная единица любого из примеров 58-60, где пептидная единица отличается тем, что вспомогательная часть, включающая одну или более нестандартных аминокислот, каждая независимо выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00576] Пример 63. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00577] Пептидная единица любого из примеров 58-60, где пептидная единица отличается тем, что каждая из первой вспомогательной части и/или второй вспомогательной части независимо выбрана из CR, HHHHHH (SEQ ID NO: 53), и RRRRRR (SEQ ID NO: 159).
[00578] Пример 64, B1A1T1A2T2A3B2
[00579] Пептидная единица любого из примеров 49 и 50, где пептидная единица отличается тем, что она дополнительно включает первую вспомогательную часть, вторую вспомогательную часть и третью вспомогательную часть; первая вспомогательная часть связана между первым В-клеточным эпитопом и первым Th-эпитопом; вторая вспомогательная часть связана между первым Th-эпитопом и вторым Th-эпитопом; и третья вспомогательная часть связана между вторым Th-эпитопом и вторым В-клеточным эпитопом.
[00580] Пример 65, Ограничение по А, включающей нестандартную аминокислоту.
[00581] Пептидная единица Примера 64, где пептидная единица отличается тем, что первая вспомогательная часть, вторая вспомогательная часть и/или третья вспомогательная часть включают одну или более нестандартных аминокислот.
[00582] Пример 66. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00583] Пептидная единица Примера 65, где вспомогательная часть, включающая одну или более нестандартных аминокислот, каждая независимо выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00584] Пример 67. Ограничение по последовательности А, не включающей нестандартную аминокислоту.
[00585] Пептидная единица Примера 64, где первая вспомогательная часть, вторая вспомогательная часть и/или третья вспомогательная часть, каждая независимо выбрана из CR, HHHHHH (SEQ ID NO: 53) и RRRRRR (SEQ ID NO: 159).
[00586] пептидная единица 7 - единица и дополнительная вспомогательная часть
[00587] Пример 68, AU и UA
[00588] пептидная единица, включающая следующее: пептидная единица любого из примеров 1-67; и дополнительная вспомогательная часть.
[00589] Пример 69. Ограничение по А, включающей нестандартную аминокислоту.
[00590] Пептидная единица Примера 67, где пептидная единица отличается тем, что дополнительная вспомогательная часть включает одну или более нестандартных аминокислот.
[00591] Пример 70. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00592] Пептидная единица примера 69, где пептидная единица отличается тем, что дополнительная вспомогательная часть выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00593] Пример 71. Ограничение по последовательности А, не включающей нестандартную аминокислоту.
[00594] Пептидная единица примера 68, где пептидная единица отличается тем, что дополнительная вспомогательная часть выбрана из CR, HHHHHH (SEQ ID NO: 53), и RRRRRR (SEQ ID NO: 159).
[00595] Пример 72, AUA
[00596] пептидная единица, включающая следующее: пептидная единица любого из примеров 1-67; и дополнительная вспомогательная часть и вторая дополнительная вспомогательная часть. В частности, пептидная единица связана между первой дополнительной вспомогательной частью и второй дополнительной вспомогательной частью.
[00597] Пример 73. Ограничение по А, включающей нестандартную аминокислоту.
[00598] Пептидная единица примера 72, где пептидная единица отличается тем, что первая дополнительная вспомогательная часть и/или вторая дополнительная вспомогательная часть включают одну или более нестандартных аминокислот.
[00599] Пример 74. Ограничение по последовательности А, включающей нестандартную аминокислоту.
[00600] Пептидная единица примера 73, где пептидная единица отличается тем, что дополнительная вспомогательная часть, включающая одну или более нестандартных аминокислот, каждая независимо выбрана из a, Z, aZ, Za, GSHHHHHHGSDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDK (SEQ ID NO: 194).
[00601] Пример 75. Ограничение по последовательности А, не включающей нестандартную аминокислоту.
[00602] Пептидная единица Примера 72, где пептидная единица отличается тем, что первая вспомогательная часть и/или вторая вспомогательная часть, каждая, независимо выбраны из CR, HHHHHH (SEQ ID NO: 53), и RRRRRR (SEQ ID NO: 159).
[00603] пептидная единица 8 - ограничение по последовательности единиц
[00604] Пример 76. Ограничение по последовательности единицы-А
[00605] Пептидная единица любого из примеров 1-8 и 68-75, где пептидная единица выбрана из следующих: RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 56); ZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF (SEQ ID NO: 57); CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 58); ZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLS (SEQ ID NO: 59); KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 60); ZaK(Cha)VAAWTLKAAaZKTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 61); RNVPPIFNDVYWIAFK(Cha)VAAWTLKAA (SEQ ID NO: 62); K(Cha)VAAWTLKAARNVPPIFNDVYWIAF (SEQ ID NO: 63); RNVPPIFNDVYK(Cha)VAAWTLKAA (SEQ ID NO: 64); PIFNDVYWIAFK(Cha)VAAWTLKAA (SEQ ID NO: 65); PPIFNDVYWK(Cha)VAAWTLKAA (SEQ ID NO: 66); RNVPPIFNDVYWIAFK(Cha)VAAWTLKAAHHHHHH (SEQ ID NO: 67); RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZGSHHHHHHGSDDDDK (SEQ ID NO: 68); GSHHHHHHGSDDDDKZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF (SEQ ID NO: 69); RNVPPIFNDVYWIAFGSHHHHHHGSDDDDKZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 70); GSHHHHHHGSDDDDKZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLS (SEQ ID NO: 71); GSHHHHHHGSDDDDKCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 72); CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZGSHHHHHHGSDDDDK (SEQ ID NO: 73); GSHHHHHHGSDDDDKZaK(Cha)VAAWTLKAAaZKTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 74); GSHHHHHHGSDDDDKKTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 75); KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZGSHHHHHHGSDDDDK (SEQ ID NO: 76); MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 77); MRGSHHHHHHGSDDDDKIVDGSHHHHHHGSDDDDKRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 78); MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZGSHHHHHHGSDDDDK (SEQ ID NO: 79); RNVPPIFNDVYWIAFILMQYIKANSKFIGI (SEQ ID NO: 80); RNVPPIFNDVYWIAFILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 81); CRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZC (SEQ ID NO: 82); RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAACR (SEQ ID NO: 161); RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXGSHHHHHHGSDDDDK (SEQ ID NO: 199); GSHHHHHHGSDDDDKXXKXVAAWTLKAAXXRNVPPIFNDVYWIAF (SEQ ID NO: 200); RNVPPIFNDVYWIAFXXKXVAAWTLKAAXX (SEQ ID NO: 204); RNVPPIFNDVYWIAFKXVAAWTLKAA (SEQ ID NO: 205); RNVPPIFNDVYWIAFKXVAAWTLKAAHHHHHH (SEQ ID NO: 206); RNVPPIFNDVYWIAFXXKXVAAWTLKAACR (SEQ ID NO: 208); RNVPPIFNDVYWIAFXXKFVAAWTLKAAXX (SEQ ID NO: 210); RNVPPIFNDVYWIAFXXKFVAAWTLKAACR (SEQ ID NO: 212); RNVPPIFNDVYWIAFCTKPTDGN (SEQ ID NO: 213); RNVPPIFNDVYWIAFLLPIFFCLW (SEQ ID NO: 214); RNVPPIFNDVYWIAFFLPSDFFPSV (SEQ ID NO: 215); RNVPPIFNDVYWIAFILMQYIKANSKFIGIHHHHHH (SEQ ID NO: 219); and RNVPPIFNDVYWIAFMDIDPYKEFGATVELLSFLPHHHHHH (SEQ ID NO: 220), в которых а обозначает D-форму аланина, Z обозначает 6-аминогексановую кислоту, (Cha) обозначает L-циклогексилаланин и X обозначает любую стандартную аминокислоту.
[00606] Пример 77, Ограничение по последовательности единицы-D
[00607] Пептидная единица любого из примеров 1-6, 14-15 и 68-75, где пептидная единица выбрана из следующих: RNVPPIFNDVYWIAF ZaK(Cha)VAAWTLKAAaZ ILMQYIKANSKFIGI (SEQ ID NO: 123); CRFRGLISLSQVYLS ZaK(Cha)VAAWTLKAAaZ ILMQYIKANSKFIGI (SEQ ID NO: 124); KTTKQSFDLSVKAQYKKNKH ZaK(Cha)VAAWTLKAAaZ ILMQYIKANSKFIGI (SEQ ID NO: 125); ILMQYIKANSKFIGI ZaK(Cha)VAAWTLKAAaZ RNVPPIFNDVYWIAF (SEQ ID NO: 126); ILMQYIKANSKFIGI ZaK(Cha)VAAWTLKAAaZ CRFRGLISLSQVYLS (SEQ ID NO: 127); ILMQYIKANSKFIGI ZaK(Cha)VAAWTLKAAaZ KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 128); PIFNDVYWIAF K(Cha)VAAWTLKAA K(Cha)VAAWTLKAA (SEQ ID NO: 129); PPIFNDVYW K(Cha)VAAWTLKAA K(Cha)VAAWTLKAA (SEQ ID NO: 130); MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGI (SEQ ID NO: 131); MRGSHHHHHHGSDDDDKIVDILMQYIKANSKFIGIZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 132); MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGI (SEQ ID NO: 133); MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSILMQYIKANSKFIGIPMGLPQSIALSSLMVAQGGGGSGGGGGGSSILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 134); RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZK(Cha)LAAFTIRAAaZ (SEQ ID NO: 135); and CRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIC (SEQ ID NO: 136), в которых а обозначает D-форму аланина, Z обозначает 6-аминогексановую кислоту и (Cha) обозначает L-циклогексилаланин.
[00608] Пример 78. Ограничение по последовательности единицы-B
[00609] Пептидная единица любого из примеров 1-6, 26-27 и 68-75, где пептидная единица выбрана из группы, состоящей из следующих: RNVPPIFNDVYWIAFRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 83), RNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ (P5: SEQ ID NO: 84), RNVPPIFNDVYWIAFKTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 85), CRFRGLISLSQVYLSRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 86), CRFRGLISLSQVYLSCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 87), CRFRGLISLSQVYLSKTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 88), KTTKQSFDLSVKAQYKKNKHRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 89), KTTKQSFDLSVKAQYKKNKHCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 90), KTTKQSFDLSVKAQYKKNKHKTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 91), RNVPPIFNDVYWIAFCRFRGLISLSQVYLSK(Cha)VAAWTLKAA (SEQ ID NO: 92), PIFNDVYWIAFGLISLSQVYLSK(Cha)VAAWTLKAA (SEQ ID NO: 93), RNVPPIFNDVYCRFRGLISLSQK(Cha)VAAWTLKAA (SEQ ID NO: 94), PIFNDVYWIAFCRFRGLISLSQK(Cha)VAAWTLKAA (SEQ ID NO: 95), PPIFNDVYWRGLISLSQVK(Cha)VAAWTLKAA (SEQ ID NO: 96), RNVPPIFNDVYWIAFCRFRGLISLSQVYLSK(Cha)VAAWTLKAAHHHHHH (SEQ ID NO: 97), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 98), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAA (SEQ ID NO: 99), RNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAA (SEQ ID NO: 100), MRGSHHHHHHGSDDDDKIVD RNVPPIFNDVYWIAF GGGGSGGGGGGSS RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAA (SEQ ID NO: 101), RNVPPIFNDVYWIAF GGGGSGGGGGGSS RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAA (SEQ ID NO: 102), RNVPPIFNDVYWIAF GGGGSGGGGGGSS RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZ (SEQ ID NO: 103), RNVPPIFNDVYWIAFRNVPPIFNDVYWIAF ILMQYIKANSKFIGI (SEQ ID NO: 104), RNVPPIFNDVYWIAFRNVPPIFNDVYWIAF ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 105), CRNVPPIFNDVYWIAFCRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZC (SEQ ID NO: 106), and RNVPPIFNDVYWIAFCRFRGLISLSQVYLSXXK(Cha)VAAWTLKAAXX (SEQ ID NO: 202), в которых а обозначает D-форму аланина, (Ch а) обозначает L-циклогексилаланины, Z обозначает 6-аминогексановую кислоту, а X обозначает любую стандартную аминокислоту.
[00610] Пример 79. Ограничение по последовательности единицы-C
[00611] Пептидная единица любого из примеров 1-6, 38-39 и 68-75, где пептидная единица выбрана из группы, состоящей из следующего: RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF (SEQ ID NO: 107); RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLS (P1: SEQ ID NO: 108); RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZKTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 109); CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF (SEQ ID NO: 110); CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLS (SEQ ID NO: 111); RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZKTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 112); KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF (SEQ ID NO: 113); KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZCRFRGLISLSQVYLS (P4: SEQ ID NO: 114); KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZKTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 115); PIFNDVYWIAFK(Cha)VAAWTLKAACRFRGLISLSQ (SEQ ID NO: 116); PPIFNDVYWK(Cha)VAAWTLKAARGLISLSQV (SEQ ID NO: 117); MRGSHHHHHHGSDDDDKIVD RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAF (SEQ ID NO: 118); MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAF GGGGSGGGGGGSS ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ GGGGSGGGGGGSSCRFRGLISLSQVYLS (SEQ ID NO: 119); RNVPPIFNDVYWIAFILMQYIKANSKFIGICRFRGLISLSQVYLS (SEQ ID NO: 120); RNVPPIFNDVYWIAFZPKYVKQNTLKLATZCRFRGLISLSQVYLS (P5: SEQ ID NO: 121); CRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZRNVPPIFNDVYWIAFC (SEQ ID NO: 122); RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAACRFRGLISLSQVYLS (SEQ ID NO: 160); RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXCRFRGLISLSQVYLS (SEQ ID NO: 198); KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXCRFRGLISLSQVYLS (SEQ ID NO: 201); RNVPPIFNDVYWIAFXPKYVKQNTLKLATXCRFRGLISLSQVYLS (SEQ ID NO: 203); RNVPPIFNDVYWIAFXXKXVAAWTLKAACRFRGLISLSQVYLS (SEQ ID NO: 207); RNVPPIFNDVYWIAFXXKFVAAWTLKAAXXCRFRGLISLSQVYLS (SEQ ID NO: 209); RNVPPIFNDVYWIAFXXKFVAAWTLKAACRFRGLISLSQVYLS (SEQ ID NO: 211); KTTKQSFDLSVKAQYKKNKHZaWPEANQVGAGAFGPGFaZCRFRGLISLSQVYLS (SEQ ID NO: 216); KTTKQSFDLSVKAQYKKNKHZaMDIDPYKEFGATVELLSFLPaZCRFRGLISLSQVYLS (SEQ ID NO: 217); and KTTKQSFDLSVKAQYKKNKHZaILMQYIKANSKFIGIPMGLPQSIALSSLMVAQaZCRFRGLISLSQVYLS (SEQ ID NO: 218), где а обозначает D-форму аланина, (Cha) обозначает L-циклогексилаланины, Z обозначает 6-аминогексановую кислоту и X обозначает любую стандартную аминокислоту.
[00612] Пример 80. Ограничение по последовательности единицы-E
[00613] Пептидная единица любого из примеров 1-6, 49-50 и 68-75, где пептидная единица выбрана из группы, состоящей из следующих: RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIRNVPPIFNDVYWIAF (SEQ ID NO: 137), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGICRFRGLISLSQVYLS (SEQ ID NO: 138), RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIKTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 139), CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIRNVPPIFNDVYWIAF (SEQ ID NO: 140), CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGICRFRGLISLSQVYLS (SEQ ID NO: 141), CRFRGLISLSQVYLSZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIKTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 142), KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIRNVPPIFNDVYWIAF (SEQ ID NO: 143), KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGICRFRGLISLSQVYLS (SEQ ID NO: 144), KTTKQSFDLSVKAQYKKNKHZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIKTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 145), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIRNVPPIFNDVYWIAF (SEQ ID NO: 146), MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSILMQYIKANSKFIGIPMGLPQSIALSSLMVAQILMQYIKANSKFIGIPMGLPQSIALSSLMVAQGGGGSGGGGGGSSCRFRGLISLSQVYLS (SEQ ID NO: 147), PIFNDVYWIAFK(Cha)VAAWTLKAAK(Cha)VAAWTLKAACRFRGLISLSQ (SEQ ID NO: 148), PPIFNDVYWK(Cha)VAAWTLKAAK(Cha)VAAWTLKAARGLISLSQV (SEQ ID NO: 149), and CRNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAAaZILMQYIKANSKFIGIRNVPPIFNDVYWIAFC (SEQ ID NO: 150), в которых а обозначает D-форму аланина, (Cha) обозначает L-циклогексилаланины и Z обозначает 6-аминогексановую кислоту.
[00614] пептидная единица 9 - формула
[00615] Пример 81. Высшая концепция формулы единицы-A
[00616] Пептидная единица любого из примеров 1-2, где пептидная единица представлена следующей [формулой A] или [формулой A']:
[00617] [Формула А] A1-B-A2-T-A3
[00618] [Формула А'] A1-T-A2-B-A3
[00619] в которой пептидная единица представляет собой единицу, которая может индуцировать гуморальный иммунитет путем распознавания CD4+Т-клетками и имеет длину, соответствующую 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру, 32-меру, 33-меру, 34-меру, 35-меру, 36-меру, 37-меру, 38-меру, 39-меру, 40-меру, 41-меру, 42-меру, 43-меру, 44-меру, 45-меру, 46-меру, 47-меру, 48-меру, 49-меру, 50-меру, 51-меру, 52-меру, 53-меру, 54-меру, 55-меру, 56-меру, 57-меру, 58-меру, 59-меру, 60-меру, 61-меру, 62-меру, 63-меру, 64-меру, 65-меру, 66-меру, 67-меру, 68-меру, 69-меру, 70-меру или 71-меру;
[00620] в которых А1 является первой вспомогательной частью или отсутствует; где первая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00621] А2 представляет собой вторую вспомогательную часть или отсутствует; где вторая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00622] А3 представляет собой третью вспомогательную часть или отсутствует; где третья вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00623] В, который представляет собой В-клеточный эпитоп, представляет собой фрагмент аполипопротеина В-100 или мимотоп аполипопротеина В-100 и может индуцировать антитела, нацеленные на аполипопротеин В-100;
[00624] Т, который представляет собой Th-эпитоп, может распознаваться CD4+Т-клетками и имеет длину, соответствующую 8-меру, 9-меру, 10-меру, 11-меру, 12-меру, 13-меру, 14-меру, 15-меру, 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру или 32-меру.
[00625] Пример 82. Комбинация последовательностей единицы-А.
[00626] В примере 81
[00627] каждый из A1, A2 и A3 может быть независимо выбран из группы, состоящей из a, Z, aZ, Za, RN, AF, CR, LS, KT, KH, RF, DP, SV, GL, ZRNV (SEQ ID NO: 36), aZRN (SEQ ID NO: 37), IAFZ (SEQ ID NO: 38), AFZa (SEQ ID NO: 39), RNVP (SEQ ID NO: 40), WIAF (SEQ ID NO: 41), ZCRF (SEQ ID NO: 42), aZCR (SEQ ID NO: 43), YLSZ (SEQ ID NO: 44), LSZa (SEQ ID NO: 45), CRFR (SEQ ID NO: 46), VYLS (SEQ ID NO: 47), ZKTT (SEQ ID NO: 48), aZKT (SEQ ID NO: 49), NKHZ (SEQ ID NO: 50), KHZa (SEQ ID NO: 51), GSHHHHHHGSDDDDK (SEQ ID NO: 52), HHHHHH (SEQ ID NO: 53), MRGSHHHHHHGSDDDDKIVD (SEQ ID NO: 54), GGGGSGGGGGGSS (SEQ ID NO: 55), RRRRRR (SEQ ID NO: 159), GSHHHHHHGSDDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDDK (SEQ ID NO: 194) или может отсутствовать, в которых а обозначает D-форму аланина, а Z обозначает 6-аминогексановую кислоту;
[00628] B содержит:
[00629] последовательность, выбранную из группы, состоящей из RNVPPIFNDVYWIAF (SEQ ID NO: 6), CRFRGLISLSQVYLS (SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8), RNVPPIFNDVY (SEQ ID NO: 9), CRFRGLISLSQ (SEQ ID NO: 10), KTTKQSFDLSVK (SEQ ID NO: 11), RNVPPIFNDVYW (SEQ ID NO: 12), CRFRGLISLSQV (SEQ ID NO: 13), KTTKQSFDLSVKAQYKK (SEQ ID NO: 14), RNVPPIFNDVYWI (SEQ ID NO: 15), CRFRGLISLSQVY (SEQ ID NO: 16), KTTKQSFDLSVKAQYKKN (SEQ ID NO: 17), PIFNDVYWIAF (SEQ ID NO: 18), GLISLSQVYLS (SEQ ID NO: 19), QSFDLSVKAQYKKNKH (SEQ ID NO: 20), PPIFNDVYWIAF (SEQ ID NO: 21), RGLISLSQVYLS (SEQ ID NO: 22), KQSFDLSVKAQYKKNKH (SEQ ID NO: 23), VPPIFNDVYWIAF (SEQ ID NO: 24), FRGLISLSQVYLS (SEQ ID NO: 25), TKQSFDLSVKAQYKKNKH (SEQ ID NO: 26), NVPPIFNDVYWIA (SEQ ID NO: 27), RFRGLISLSQVYL (SEQ ID NO: 28), TKQSFDLSVKAQYKKN (SEQ ID NO: 29), VPPIFNDVYWI (SEQ ID NO: 30), FRGLISLSQVY (SEQ ID NO: 31), TKQSFDLSVKAQYKKN (SEQ ID NO: 32), PPIFNDVYW (SEQ ID NO: 33), RGLISLSQV (SEQ ID NO: 34), KQSFDLSVKAQYKK (SEQ ID NO: 35), RFRGLISLSQVYLDP (SEQ ID NO: 221), и SVCGCPVGHHDVVGL (SEQ ID NO: 222);
[00630] эпитоп, который включен в любую из последовательностей SEQ ID NO: 6 - SEQ ID NO: 35 и SEQ ID NO: 221 - SEQ ID NO: 222; или
[00631] последовательность, которая соответствует 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88% или более, 89% или более, 90% или более, 91% или более, 92% или более, 93% или более, 94% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более, или 100% или более, любой из последовательностей SEQ ID NO: 6 - SEQ ID NO: 35 и SEQ ID NO: 221 - SEQ ID NO: 222;
[00632] в которых а обозначает D-форму аланина, а Z обозначает 6-аминогексановую кислоту;
[00633] Т выбирают из группы, состоящей из следующего:
[00634] последовательности, выбранной из группы, состоящей из K(Cha)VAAWTLKAA (SEQ ID NO: 1), PKYVKQNTLKLAT (SEQ ID NO: 2), ILMQYIKANSKFIGI (SEQ ID NO: 3), QSIALSSLMVAQAIP (SEQ ID NO: 4), ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 5), PLGFFPDHQL (SEQ ID NO: 162), WPEANQVGAGAFGPGF (SEQ ID NO: 163), MQWNSTALHQALQDP (SEQ ID NO: 164), MQWNSTTFHQTLQDPRVRGLYFPAGG (SEQ ID NO: 165), FFLLTRILTI (SEQ ID NO: 166), FFLLTRILTIPQSLD (SEQ ID NO: 167), TSLNFLGGTTVCLGQ (SEQ ID NO: 168), QSPTSNHSPTSCPPIC (SEQ ID NO: 169), IIFLFILLLCLIFLLVLLD (SEQ ID NO: 170), CTTPAQGNSMFPSC (SEQ ID NO: 171), CTKPTDGN (SEQ ID NO: 172), WASVRFSW (SEQ ID NO: 173), LLPIFFCLW (SEQ ID NO: 174), MDIDPYKEFGATVELLSFLP (SEQ ID NO: 175), FLPSDFFPSV (SEQ ID NO: 176), RDLLDTASALYREALESPEH (SEQ ID NO: 177), PHHTALRQAILCWGELMTLA (SEQ ID NO: 178), GRETVIEYLVSFGVW (SEQ ID NO: 179), EYLVSFGVWIRTPPA (SEQ ID NO: 180), VSFGVWIRTPPAYRPPNAPI (SEQ ID NO: 181), TVVRRRGRSP (SEQ ID NO: 182), VGPLTVNEKRRLKLI (SEQ ID NO: 183), RHYLHTLWKAGILYK (SEQ ID NO: 184), ESRLVVDFSQFSRGN (SEQ ID NO: 185), LQSLTNLLSSNLSWL (SEQ ID NO: 186), SSNLSWLSLDVSAAF (SEQ ID NO: 187), LHLYSHPIILGFRKI (SEQ ID NO: 188), KQCFRKLPVNRPIDW (SEQ ID NO: 189), LCQVFADATPTGWGL (SEQ ID NO: 190), AANWILRGTSFVYVP (SEQ ID NO: 191), EIRLKVFVLGGCRHK (SEQ ID NO: 192), KFVAAWTLKAA (SEQ ID NO: 195), KYVAAWTLKAA (SEQ ID NO: 196), DIEKKIAKMEKASSVFNVVNS (SEQ ID NO: 223), YSGPLKAEIAQRLEDV (SEQ ID NO: 224), K(Cha)VKANTLKAA (SEQ ID NO: 225), K(Cha)VKANTLKAA (SEQ ID NO: 226), K(Cha)VKAWTLKAA (SEQ ID NO: 227), K(Cha)VKAWTLKAA (SEQ ID NO: 228), K(Cha)VWANTLKAA (SEQ ID NO: 229), K(Cha)VWANTLKAA (SEQ ID NO: 230), K(Cha)VWAYTLKAA (SEQ ID NO: 231), K(Cha)VWAVTLKAA (SEQ ID NO: 232), K(Cha)VYAWTLKAA (SEQ ID NO: 233), K(Cha)VYAWTLKAA (SEQ ID NO: 234), R(Cha)VRANTLKAA (SEQ ID NO: 235), K(Cha)VKAHTLKAA (SEQ ID NO: 236), K(Cha)VKAHTLKAA (SEQ ID NO: 237), K(Cha)VAANTLKAA (SEQ ID NO: 238), K(Cha)VAANTLKAA (SEQ ID NO: 239), K(Cha)VAAYTLKAA (SEQ ID NO: 240), K(Cha)VAAYTLKAA (SEQ ID NO: 241), K(Cha)VAAWTLKAA (SEQ ID NO: 242), K(Cha)VAAKTLKAA (SEQ ID NO: 243), K(Cha)VAAHTLKAA (SEQ ID NO: 244), K(Cha)VAAATLKAA (SEQ ID NO: 245), K(Cha)VAAWTLKAA (SEQ ID NO: 246), и K(Cha)VMAATLKAA (SEQ ID NO: 247);
[00635] последовательности, представленной следующей [Формулой I] или [Формулой II];
[00636] [Формула I] N-Lys-X1-X2-Ala-Ala-X3-Thr-X4-X5-Ala-Ala-C
[00637] в которой X1 представляет собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин;
[00638] X2 представляет собой гидрофобную аминокислоту, или лейцин (Leu), или изолейцин (Ile);
[00639] X3 представляет собой ароматическую или циклическую аминокислоту или фенилаланин (Phe), тирозин (Tyr) или гистидин (His);
[00640] X4 представляет собой алифатическую аминокислоту с длинной цепью, или изолейцин (Ile), или валин (Val); и
[00641] X5 представляет собой заряженную аминокислоту или аргинин (Arg), лейцин (Leu), аспарагиновую кислоту (Asp), глутамин (Gln) или глицин (Gly);
[00642] [Формула II] N-Lys-X1-Val-X2-Ala-X3-Thr-Leu-Lys-Ala-Ala-C
[00643] в которой X1 представляет собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин;
[00644] X2 представляет собой лизин (Lys), триптофан (Trp), тирозин (Tyr), аргинин (Arg), аланин (Ala) или метионин (Met); и
[00645] X3 представляет собой аспарагин (Asn), триптофан (Trp), тирозин (Tyr), валин (Val), гистидин (His), лизин (Lys) или аланин (Ala); и
[00646] последовательности, которая соответствует 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88% или более, 89% или более, 90% или более, 91% или более, 92% или более, 93% или более, 94% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более, или 100% или более любой из последовательностей, представленных SEQ ID NO: 1 - SEQ ID NO: 5, SEQ ID NO: 162 - SEQ ID NO: 192, SEQ ID NO: 195 - SEQ ID NO: 196, SEQ ID NO: 223 - SEQ ID NO: 247 и [формулой I] или [формулой II];
[00647] в которой (Cha) обозначает L-циклогексилаланины, а X обозначает любую стандартную аминокислоту.
[00648] Пример 83. Высшая концепция формулы единицы-B
[00649] Пептидная единица любого из примеров 1 и 2, где пептидная единица представлена следующей [формулой B] или [формулой B']:
[00650] [Формула B] A1-B1-A2-B2-A3-T-A4
[00651] [Формула B'] A1-T-A2-B1-A3-B2-A4
[00652] в которой пептидная единица отличается тем, что она может индуцировать гуморальный иммунитет за счет распознавания CD4+Т-клетками и имеет длину, соответствующую 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру, 32-меру, 33-меру, 34-меру, 5-меру, 36-меру, 37-меру, 38-меру, 39-меру, 40-меру, 41-меру, 42-меру, 43-меру, 44-меру, 45-меру, 46-меру, 47-меру, 48-меру, 49-меру, 50-меру, 51-меру, 52-меру, 53-меру, 54-меру, 55-меру, 56-меру, 57-меру, 58-меру, 59-меру, 60-меру, 61-меру, 62-меру, 63-меру, 64-меру, 65-меру, 66-меру, 67-меру, 68-меру, 69-меру, 70-меру или 71-меру;
[00653] в которых А1 является первой вспомогательной частью или отсутствует; где первая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00654] А2 представляет собой вторую вспомогательную часть или отсутствует; где вторая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00655] А3 представляет собой третью вспомогательную часть или отсутствует; где третья вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00656] А4 представляет собой четвертую вспомогательную часть или отсутствует; где четвертая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00657] В1, который представляет собой В-клеточный эпитоп, представляет собой фрагмент аполипопротеина В-100 или мимотоп аполипопротеина В-100 и может индуцировать антитела, нацеленные на аполипопротеин В-100;
[00658] В2, который представляет собой В-клеточный эпитоп, представляет собой фрагмент аполипопротеина В-100 или мимотоп аполипопротеина В-100 и может индуцировать антитела, нацеленные на аполипопротеин В-100; и
[00659] Т, который представляет собой Th-эпитоп, может распознаваться CD4+Т-клетками и имеет длину, соответствующую 8-меру, 9-меру, 10-меру, 11-меру, 12-меру, 13-меру, 14-меру, 15-меру, 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру или 32-меру.
[00660] Пример 84. Комбинация последовательностей единицы-B.
[00661] В примере 83
[00662] каждый из A1, A2 и A3 независимо выбран из группы, состоящей из a, Z, aZ, Za, RN, AF, CR, LS, KT, KH, RF, DP, SV, GL, ZRNV (SEQ ID NO: 36), aZRN (SEQ ID NO: 37), IAFZ (SEQ ID NO: 38), AFZa (SEQ ID NO: 39), RNVP (SEQ ID NO: 40), WIAF (SEQ ID NO: 41), ZCRF (SEQ ID NO: 42), aZCR (SEQ ID NO: 43), YLSZ (SEQ ID NO: 44), LSZa (SEQ ID NO: 45), CRFR (SEQ ID NO: 46), VYLS (SEQ ID NO: 47), ZKTT (SEQ ID NO: 48), aZKT (SEQ ID NO: 49), NKHZ (SEQ ID NO: 50), KHZa (SEQ ID NO: 51), GSHHHHHHGSDDDDK (SEQ ID NO: 52), HHHHHH (SEQ ID NO: 53), MRGSHHHHHHGSDDDDKIVD (SEQ ID NO: 54), GGGGSGGGGGGSS (SEQ ID NO: 55), RRRRRR (SEQ ID NO: 159), GSHHHHHHGSDDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDDK (SEQ ID NO: 194) или отсутствует; в которой а обозначает D-форму аланина, а Z обозначает 6-аминогексановую кислоту;
[00663] B1 и B2 независимо друг от друга,
[00664] каждый выбран из группы, состоящей из RNVPPIFNDVYWIAF (SEQ ID NO: 6), CRFRGLISLSQVYLS (SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8), RNVPPIFNDVY (SEQ ID NO: 9), CRFRGLISLSQ (SEQ ID NO: 10), KTTKQSFDLSVK (SEQ ID NO: 11), RNVPPIFNDVYW (SEQ ID NO: 12), CRFRGLISLSQV (SEQ ID NO: 13), KTTKQSFDLSVKAQYKK (SEQ ID NO: 14), RNVPPIFNDVYWI (SEQ ID NO: 15), CRFRGLISLSQVY (SEQ ID NO: 16), KTTKQSFDLSVKAQYKKN (SEQ ID NO: 17), PIFNDVYWIAF (SEQ ID NO: 18), GLISLSQVYLS (SEQ ID NO: 19), QSFDLSVKAQYKKNKH (SEQ ID NO: 20), PPIFNDVYWIAF (SEQ ID NO: 21), RGLISLSQVYLS (SEQ ID NO: 22), KQSFDLSVKAQYKKNKH (SEQ ID NO: 23), VPPIFNDVYWIAF (SEQ ID NO: 24), FRGLISLSQVYLS (SEQ ID NO: 25), TKQSFDLSVKAQYKKNKH (SEQ ID NO: 26), NVPPIFNDVYWIA (SEQ ID NO: 27), RFRGLISLSQVYL (SEQ ID NO: 28), TKQSFDLSVKAQYKKN (SEQ ID NO: 29), VPPIFNDVYWI (SEQ ID NO: 30), FRGLISLSQVY (SEQ ID NO: 31), TKQSFDLSVKAQYKKN (SEQ ID NO: 32), PPIFNDVYW (SEQ ID NO: 33), RGLISLSQV (SEQ ID NO: 34), KQSFDLSVKAQYKK (SEQ ID NO: 35), RFRGLISLSQVYLDP (SEQ ID NO: 221), и SVCGCPVGHHDVVGL (SEQ ID NO: 222);
[00665] эпитопа, который включен в любую из последовательностей SEQ ID NO: 6 - SEQ ID NO: 35 и SEQ ID NO: 221 - SEQ ID NO: 222; или
[00666] имеет последовательности, которая соответствует 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88% или более, 89% и более, 90% и более, 91% и более, 92% и более, 93% и более, 94% и более, 95% и более, 96% и более, 97% и более, 98% и более, 99% или более или 100% или более любой из последовательностей SEQ ID NO: 6 - SEQ ID NO: 35 и от SEQ ID NO: 221 - SEQ ID NO: 222;
[00667] в которых а обозначает D-форму аланина, а Z обозначает 6-аминогексановую кислоту;
[00668] Т выбирают из группы, состоящей из следующего:
[00669] последовательности, выбранный из группы, состоящей из K(Cha)VAAWTLKAA (SEQ ID NO: 1), PKYVKQNTLKLAT (SEQ ID NO: 2), ILMQYIKANSKFIGI (SEQ ID NO: 3), QSIALSSLMVAQAIP (SEQ ID NO: 4), ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 5), PLGFFPDHQL (SEQ ID NO: 162), WPEANQVGAGAFGPGF (SEQ ID NO: 163), MQWNSTALHQALQDP (SEQ ID NO: 164), MQWNSTTFHQTLQDPRVRGLYFPAGG (SEQ ID NO: 165), FFLLTRILTI (SEQ ID NO: 166), FFLLTRILTIPQSLD (SEQ ID NO: 167), TSLNFLGGTTVCLGQ (SEQ ID NO: 168), QSPTSNHSPTSCPPIC (SEQ ID NO: 169), IIFLFILLLCLIFLLVLLD (SEQ ID NO: 170), CTTPAQGNSMFPSC (SEQ ID NO: 171), CTKPTDGN (SEQ ID NO: 172), WASVRFSW (SEQ ID NO: 173), LLPIFFCLW (SEQ ID NO: 174), MDIDPYKEFGATVELLSFLP (SEQ ID NO: 175), FLPSDFFPSV (SEQ ID NO: 176), RDLLDTASALYREALESPEH (SEQ ID NO: 177), PHHTALRQAILCWGELMTLA (SEQ ID NO: 178), GRETVIEYLVSFGVW (SEQ ID NO: 179), EYLVSFGVWIRTPPA (SEQ ID NO: 180), VSFGVWIRTPPAYRPPNAPI (SEQ ID NO: 181), TVVRRRGRSP (SEQ ID NO: 182), VGPLTVNEKRRLKLI (SEQ ID NO: 183), RHYLHTLWKAGILYK (SEQ ID NO: 184), ESRLVVDFSQFSRGN (SEQ ID NO: 185), LQSLTNLLSSNLSWL (SEQ ID NO: 186), SSNLSWLSLDVSAAF (SEQ ID NO: 187), LHLYSHPIILGFRKI (SEQ ID NO: 188), KQCFRKLPVNRPIDW (SEQ ID NO: 189), LCQVFADATPTGWGL (SEQ ID NO: 190), AANWILRGTSFVYVP (SEQ ID NO: 191), EIRLKVFVLGGCRHK (SEQ ID NO: 192), KFVAAWTLKAA (SEQ ID NO: 195), KYVAAWTLKAA (SEQ ID NO: 196), DIEKKIAKMEKASSVFNVVNS (SEQ ID NO: 223), YSGPLKAEIAQRLEDV (SEQ ID NO: 224), K(Cha)VKANTLKAA (SEQ ID NO: 225), K(Cha)VKANTLKAA (SEQ ID NO: 226), K(Cha)VKAWTLKAA (SEQ ID NO: 227), K(Cha)VKAWTLKAA (SEQ ID NO: 228), K(Cha)VWANTLKAA (SEQ ID NO: 229), K(Cha)VWANTLKAA (SEQ ID NO: 230), K(Cha)VWAYTLKAA (SEQ ID NO: 231), K(Cha)VWAVTLKAA (SEQ ID NO: 232), K(Cha)VYAWTLKAA (SEQ ID NO: 233), K(Cha)VYAWTLKAA (SEQ ID NO: 234), R(Cha)VRANTLKAA (SEQ ID NO: 235), K(Cha)VKAHTLKAA (SEQ ID NO: 236), K(Cha)VKAHTLKAA (SEQ ID NO: 237), K(Cha)VAANTLKAA (SEQ ID NO: 238), K(Cha)VAANTLKAA (SEQ ID NO: 239), K(Cha)VAAYTLKAA (SEQ ID NO: 240), K(Cha)VAAYTLKAA (SEQ ID NO: 241), K(Cha)VAAWTLKAA (SEQ ID NO: 242), K(Cha)VAAKTLKAA (SEQ ID NO: 243), K(Cha)VAAHTLKAA (SEQ ID NO: 244), K(Cha)VAAATLKAA (SEQ ID NO: 245), K(Cha)VAAWTLKAA (SEQ ID NO: 246), и K(Cha)VMAATLKAA (SEQ ID NO: 247);
[00670] последовательности, представленной следующей [Формулой I] или [Формулой II]:
[00671] [Формула I] N-Lys-X1-X2-Ala-Ala-X3-Thr-X4-X5-Ala-Ala-C
[00672] в которой X1 представляет собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин;
[00673] X2 представляет собой гидрофобную аминокислоту, или лейцин (Leu), или изолейцин (Ile);
[00674] X3 представляет собой ароматическую или циклическую аминокислоту или фенилаланин (Phe), тирозин (Tyr) или гистидин (His);
[00675] X4 представляет собой алифатическую аминокислоту с длинной цепью, или изолейцин (Ile), или валин (Val); и
[00676] X5 представляет собой заряженную аминокислоту или аргинин (Arg), лейцин (Leu), аспарагиновую кислоту (Asp), глутамин (Gln) или глицин (Gly);
[00677] [Формула II] N-Lys-X1-Val-X2-Ala-X3-Thr-Leu-Lys-Ala-Ala-C
[00678] в которой X1 представляет собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин;
[00679] X2 представляет собой лизин (Lys), триптофан (Trp), тирозин (Tyr), аргинин (Arg), аланин (Ala) или метионин (Met); и
[00680] X3 представляет собой аспарагин (Asn), триптофан (Trp), тирозин (Tyr), валин (Val), гистидин (His), лизин (Lys) или аланин (Ala); и
[00681] последовательности, которая соответствует 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88% или более, 89% или более, 90% или более, 91% или более, 92% или более, 93% или более, 94% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более, или 100% или более любой из последовательностей, представленных SEQ ID NO: 1 - SEQ ID NO: 5, SEQ ID NO: 162 - SEQ ID NO: 192, SEQ ID NO: 195 - SEQ ID NO: 196, SEQ ID NO: 223 - SEQ ID NO: 247 и [формулой I] или [формулой II];
[00682] в которой (Cha) обозначает L-циклогексилаланины, а X обозначает любую стандартную аминокислоту.
[00683] Пример 85. Высшая концепция формулы единицы-C
[00684] Пептидная единица любого из примеров 1-2, где пептидная единица представлена следующей [формулой C]:
[00685] [Формула C] A1-B1-A2-T-A3-B2-A4
[00686] в которой пептидная единица отличается тем, что она может индуцировать гуморальный иммунитет за счет распознавания CD4+Т-клетками и имеет длину, соответствующую 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру, 32-меру, 33-меру, 34-меру, 5-меру, 36-меру, 37-меру, 38-меру, 39-меру, 40-меру, 41-меру, 42-меру, 43-меру, 44-меру, 45-меру, 46-меру, 47-меру, 48-меру, 49-меру, 50-меру, 51-меру, 52-меру, 53-меру, 54-меру, 55-меру, 56-меру, 57-меру, 58-меру, 59-меру, 60-меру, 61-меру, 62-меру, 63-меру, 64-меру, 65-меру, 66-меру, 67-меру, 68-меру, 69-меру, 70-меру или 71-меру;
[00687] в которых А1 является первой вспомогательной частью или отсутствует; где первая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00688] А2 представляет собой вторую вспомогательную часть или отсутствует; где вторая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00689] А3 представляет собой третью вспомогательную часть или отсутствует; где третья вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00690] А4 представляет собой четвертую вспомогательную часть или отсутствует; где четвертая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00691] В1, который представляет собой В-клеточный эпитоп, представляет собой фрагмент аполипопротеина В-100 или мимотоп аполипопротеина В-100 и может индуцировать антитела, нацеленные на аполипопротеин В-100;
[00692] В2, который представляет собой В-клеточный эпитоп, представляет собой фрагмент аполипопротеина В-100 или мимотоп аполипопротеина В-100 и может индуцировать антитела, нацеленные на аполипопротеин В-100; и
[00693] Т, который представляет собой Th-эпитоп, может распознаваться CD4+Т-клетками и имеет длину, соответствующую 8-меру, 9-меру, 10-меру, 11-меру, 12-меру, 13-меру, 14-меру, 15-меру, 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру или 32-меру.
[00694] Пример 86. Комбинация последовательностей единицы-C
[00695] В примере 85
[00696] A1, A2, A3 и A4, каждый независимо выбран из группы, состоящей из a, Z, aZ, Za, RN, AF, CR, LS, KT, KH, RF, DP, SV, GL, ZRNV (SEQ ID NO: 36), aZRN (SEQ ID NO: 37), IAFZ (SEQ ID NO: 38), AFZa (SEQ ID NO: 39), RNVP (SEQ ID NO: 40), WIAF (SEQ ID NO: 41), ZCRF (SEQ ID NO: 42), aZCR (SEQ ID NO: 43), YLSZ (SEQ ID NO: 44), LSZa (SEQ ID NO: 45), CRFR (SEQ ID NO: 46), VYLS (SEQ ID NO: 47), ZKTT (SEQ ID NO: 48), aZKT (SEQ ID NO: 49), NKHZ (SEQ ID NO: 50), KHZa (SEQ ID NO: 51), GSHHHHHHGSDDDDK (SEQ ID NO: 52), HHHHHH (SEQ ID NO: 53), MRGSHHHHHHGSDDDDKIVD (SEQ ID NO: 54), GGGGSGGGGGGSS (SEQ ID NO: 55), RRRRRR (SEQ ID NO: 159), GSHHHHHHGSDDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDDK (SEQ ID NO: 194), либо отсутствует; в которых а обозначает D-форму аланина, а Z обозначает 6-аминогексановую кислоту;
[00697] B1 и B2, каждый независимо содержат последовательность, выбранную из группы, состоящей из:
[00698] последовательности, выбранной из группы, состоящей из RNVPPIFNDVYWIAF (SEQ ID NO: 6), CRFRGLISLSQVYLS (SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8), RNVPPIFNDVY (SEQ ID NO: 9), CRFRGLISLSQ (SEQ ID NO: 10), KTTKQSFDLSVK (SEQ ID NO: 11), RNVPPIFNDVYW (SEQ ID NO: 12), CRFRGLISLSQV (SEQ ID NO: 13), KTTKQSFDLSVKAQYKK (SEQ ID NO: 14), RNVPPIFNDVYWI (SEQ ID NO: 15), CRFRGLISLSQVY (SEQ ID NO: 16), KTTKQSFDLSVKAQYKKN (SEQ ID NO: 17), PIFNDVYWIAF (SEQ ID NO: 18), GLISLSQVYLS (SEQ ID NO: 19), QSFDLSVKAQYKKNKH (SEQ ID NO: 20), PPIFNDVYWIAF (SEQ ID NO: 21), RGLISLSQVYLS (SEQ ID NO: 22), KQSFDLSVKAQYKKNKH (SEQ ID NO: 23), VPPIFNDVYWIAF (SEQ ID NO: 24), FRGLISLSQVYLS (SEQ ID NO: 25), TKQSFDLSVKAQYKKNKH (SEQ ID NO: 26), NVPPIFNDVYWIA (SEQ ID NO: 27), RFRGLISLSQVYL (SEQ ID NO: 28), TKQSFDLSVKAQYKKN (SEQ ID NO: 29), VPPIFNDVYWI (SEQ ID NO: 30), FRGLISLSQVY (SEQ ID NO: 31), TKQSFDLSVKAQYKKN (SEQ ID NO: 32), PPIFNDVYW (SEQ ID NO: 33), RGLISLSQV (SEQ ID NO: 34), KQSFDLSVKAQYKK (SEQ ID NO: 35), RFRGLISLSQVYLDP (SEQ ID NO: 221), и SVCGCPVGHHDVVGL (SEQ ID NO: 222);
[00699] эпитопа, который включен в любую из последовательностей SEQ ID NO: 6 - SEQ ID NO: 35 и SEQ ID NO: 221 - SEQ ID NO: 222; или
[00700] последовательности, которая соответствует 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88% или более, 89% или более, 90% или более, 91% или более, 92% или более, 93% или более, 94% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более, или 100% или более любой из последовательностей, представленных SEQ ID NO: 6 - SEQ ID NO: 35 и от SEQ ID NO: 221 - SEQ ID NO: 222;
[00701] в которой «а» обозначает D-форму аланина, и «Z» обозначает 6-аминогексановую кислоту;
[00702] Т выбирают из группы, состоящей из следующего:
[00703] последовательности, выбранной из группы, состоящей из K(Cha)VAAWTLKAA (SEQ ID NO: 1), PKYVKQNTLKLAT (SEQ ID NO: 2), ILMQYIKANSKFIGI (SEQ ID NO: 3), QSIALSSLMVAQAIP (SEQ ID NO: 4), ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 5), PLGFFPDHQL (SEQ ID NO: 162), WPEANQVGAGAFGPGF (SEQ ID NO: 163), MQWNSTALHQALQDP (SEQ ID NO: 164), MQWNSTTFHQTLQDPRVRGLYFPAGG (SEQ ID NO: 165), FFLLTRILTI (SEQ ID NO: 166), FFLLTRILTIPQSLD (SEQ ID NO: 167), TSLNFLGGTTVCLGQ (SEQ ID NO: 168), QSPTSNHSPTSCPPIC (SEQ ID NO: 169), IIFLFILLLCLIFLLVLLD (SEQ ID NO: 170), CTTPAQGNSMFPSC (SEQ ID NO: 171), CTKPTDGN (SEQ ID NO: 172), WASVRFSW (SEQ ID NO: 173), LLPIFFCLW (SEQ ID NO: 174), MDIDPYKEFGATVELLSFLP (SEQ ID NO: 175), FLPSDFFPSV (SEQ ID NO: 176), RDLLDTASALYREALESPEH (SEQ ID NO: 177), PHHTALRQAILCWGELMTLA (SEQ ID NO: 178), GRETVIEYLVSFGVW (SEQ ID NO: 179), EYLVSFGVWIRTPPA (SEQ ID NO: 180), VSFGVWIRTPPAYRPPNAPI (SEQ ID NO: 181), TVVRRRGRSP (SEQ ID NO: 182), VGPLTVNEKRRLKLI (SEQ ID NO: 183), RHYLHTLWKAGILYK (SEQ ID NO: 184), ESRLVVDFSQFSRGN (SEQ ID NO: 185), LQSLTNLLSSNLSWL (SEQ ID NO: 186), SSNLSWLSLDVSAAF (SEQ ID NO: 187), LHLYSHPIILGFRKI (SEQ ID NO: 188), KQCFRKLPVNRPIDW (SEQ ID NO: 189), LCQVFADATPTGWGL (SEQ ID NO: 190), AANWILRGTSFVYVP (SEQ ID NO: 191), EIRLKVFVLGGCRHK (SEQ ID NO: 192), KFVAAWTLKAA (SEQ ID NO: 195), KYVAAWTLKAA (SEQ ID NO: 196), DIEKKIAKMEKASSVFNVVNS (SEQ ID NO: 223), YSGPLKAEIAQRLEDV (SEQ ID NO: 224), K(Cha)VKANTLKAA (SEQ ID NO: 225), K(Cha)VKANTLKAA (SEQ ID NO: 226), K(Cha)VKAWTLKAA (SEQ ID NO: 227), K(Cha)VKAWTLKAA (SEQ ID NO: 228), K(Cha)VWANTLKAA (SEQ ID NO: 229), K(Cha)VWANTLKAA (SEQ ID NO: 230), K(Cha)VWAYTLKAA (SEQ ID NO: 231), K(Cha)VWAVTLKAA (SEQ ID NO: 232), K(Cha)VYAWTLKAA (SEQ ID NO: 233), K(Cha)VYAWTLKAA (SEQ ID NO: 234), R(Cha)VRANTLKAA (SEQ ID NO: 235), K(Cha)VKAHTLKAA (SEQ ID NO: 236), K(Cha)VKAHTLKAA (SEQ ID NO: 237), K(Cha)VAANTLKAA (SEQ ID NO: 238), K(Cha)VAANTLKAA (SEQ ID NO: 239), K(Cha)VAAYTLKAA (SEQ ID NO: 240), K(Cha)VAAYTLKAA (SEQ ID NO: 241), K(Cha)VAAWTLKAA (SEQ ID NO: 242), K(Cha)VAAKTLKAA (SEQ ID NO: 243), K(Cha)VAAHTLKAA (SEQ ID NO: 244), K(Cha)VAAATLKAA (SEQ ID NO: 245), K(Cha)VAAWTLKAA (SEQ ID NO: 246), и K(Cha)VMAATLKAA (SEQ ID NO: 247);
[00704] последовательности, представленной следующей [Формулой I] или [Формулой II]:
[00705] [Формула I] N-Lys-X1-X2-Ala-Ala-X3-Thr-X4-X5-Ala-Ala-C
[00706] в которой X1 представляет собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин;
[00707] X2 представляет собой гидрофобную аминокислоту, или лейцин (Leu), или изолейцин (Ile);
[00708] X3 представляет собой ароматическую или циклическую аминокислоту или фенилаланин (Phe), тирозин (Tyr) или гистидин (His);
[00709] X4 представляет собой алифатическую аминокислоту с длинной цепью, или изолейцин (Ile), или валин (Val); и
[00710] X5 представляет собой заряженную аминокислоту или аргинин (Arg), лейцин (Leu), аспарагиновую кислоту (Asp), глутамин (Gln) или глицин (Gly);
[00711] [Формула II] N-Lys-X1-Val-X2-Ala-X3-Thr-Leu-Lys-Ala-Ala-C
[00712] в которой X1 представляет собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин;
[00713] X2 представляет собой лизин (Lys), триптофан (Trp), тирозин (Tyr), аргинин (Arg), аланин (Ala) или метионин (Met); и
[00714] X3 представляет собой аспарагин (Asn), триптофан (Trp), тирозин (Tyr), валин (Val), гистидин (His), лизин (Lys) или аланин (Ala); и
[00715] последовательности, которая соответствует 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88% или более, 89% или более, 90% или более, 91% или более, 92% или более, 93% или более, 94% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более, или 100% или более любой из последовательностей, представленных SEQ ID NO: 1 - SEQ ID NO: 5, SEQ ID NO: 162 - SEQ ID NO: 192, SEQ ID NO: 195 - SEQ ID NO: 196, SEQ ID NO: 223 - SEQ ID NO: 247 и [формулой I] или [формулой II];
[00716] в которой (Cha) обозначает L-циклогексилаланины, а X обозначает любую стандартную аминокислоту.
[00717] Пример 87. Высшая концепция формулы единицы-D
[00718] Пептидная единица любого из примеров 1 и 2, где пептидная единица представлена следующей [Формулой I] или [Формулой II]:
[00719] [Формула D] A1-B-A2-T1-A3-T2-A4
[00720] [Формула D'] A1-T1-A2-T2-A3-B-A4
[00721] в которой пептидная единица отличается тем, что она может индуцировать гуморальный иммунитет за счет распознавания CD4+Т-клетками и имеет длину, соответствующую 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру, 32-меру, 33-меру, 34-меру, 5-меру, 36-меру, 37-меру, 38-меру, 39-меру, 40-меру, 41-меру, 42-меру, 43-меру, 44-меру, 45-меру, 46-меру, 47-меру, 48-меру, 49-меру, 50-меру, 51-меру, 52-меру, 53-меру, 54-меру, 55-меру, 56-меру, 57-меру, 58-меру, 59-меру, 60-меру, 61-меру, 62-меру, 63-меру, 64-меру, 65-меру, 66-меру, 67-меру, 68-меру, 69-меру, 70-меру или 71-меру;
[00722] в которых А1 является первой вспомогательной частью или отсутствует; где первая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00723] А2 представляет собой вторую вспомогательную часть или отсутствует; где вторая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и необязательно может содержать нестандартную аминокислоту;
[00724] А3 представляет собой третью вспомогательную часть или отсутствует; где третья вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00725] А4 представляет собой четвертую вспомогательную часть или отсутствует; где четвертая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00726] В, который представляет собой В-клеточный эпитоп, представляет собой фрагмент аполипопротеина В-100 или мимотоп аполипопротеина В-100 и может индуцировать антитела, нацеленные на аполипопротеин В-100;
[00727] Т1, который представляет собой Th-эпитоп, может распознаваться CD4+Т-клетками и имеет длину, соответствующую 8-меру, 9-меру, 10-меру, 11-меру, 12-меру, 13-меру, 14-меру, 15-меру, 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру или 32-меру; и
[00728] Т2, который представляет собой Th-эпитоп, может распознаваться CD4+Т-клетками и имеет длину, соответствующую 8 меру, 9 меру, 10 меру, 11 меру, 12 меру, 13 меру, 14 меру, 15 меру, 16 меру, 17 меру, 18 меру, 19 меру, 20 меру, 21 меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру или 32-меру.
[00729] Пример 88. Комбинация последовательностей Единицы-D
[00730] В примере 87
[00731] A1, A2, A3 и A4, каждый независимо выбран из группы, состоящей из a, Z, aZ, Za, RN, AF, CR, LS, KT, KH, RF, DP, SV, GL, ZRNV (SEQ ID NO: 36), aZRN (SEQ ID NO: 37), IAFZ (SEQ ID NO: 38), AFZa (SEQ ID NO: 39), RNVP (SEQ ID NO: 40), WIAF (SEQ ID NO: 41), ZCRF (SEQ ID NO: 42), aZCR (SEQ ID NO: 43), YLSZ (SEQ ID NO: 44), LSZa (SEQ ID NO: 45), CRFR (SEQ ID NO: 46), VYLS (SEQ ID NO: 47), ZKTT (SEQ ID NO: 48), aZKT (SEQ ID NO: 49), NKHZ (SEQ ID NO: 50), KHZa (SEQ ID NO: 51), GSHHHHHHGSDDDDK (SEQ ID NO: 52), HHHHHH (SEQ ID NO: 53), MRGSHHHHHHGSDDDDKIVD (SEQ ID NO: 54), GGGGSGGGGGGSS (SEQ ID NO: 55), RRRRRR (SEQ ID NO: 159), GSHHHHHHGSDDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDDK (SEQ ID NO: 194), либо отсутствует; в которой а обозначает D-форму аланина, а Z обозначает 6-аминогексановую кислоту;
[00732] B содержит:
[00733] последовательность, выбранную из группы, состоящей из RNVPPIFNDVYWIAF (SEQ ID NO: 6), CRFRGLISLSQVYLS (SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8), RNVPPIFNDVY (SEQ ID NO: 9), CRFRGLISLSQ (SEQ ID NO: 10), KTTKQSFDLSVK (SEQ ID NO: 11), RNVPPIFNDVYW (SEQ ID NO: 12), CRFRGLISLSQV (SEQ ID NO: 13), KTTKQSFDLSVKAQYKK (SEQ ID NO: 14), RNVPPIFNDVYWI (SEQ ID NO: 15), CRFRGLISLSQVY (SEQ ID NO: 16), KTTKQSFDLSVKAQYKKN (SEQ ID NO: 17), PIFNDVYWIAF (SEQ ID NO: 18), GLISLSQVYLS (SEQ ID NO: 19), QSFDLSVKAQYKKNKH (SEQ ID NO: 20), PPIFNDVYWIAF (SEQ ID NO: 21), RGLISLSQVYLS (SEQ ID NO: 22), KQSFDLSVKAQYKKNKH (SEQ ID NO: 23), VPPIFNDVYWIAF (SEQ ID NO: 24), FRGLISLSQVYLS (SEQ ID NO: 25), TKQSFDLSVKAQYKKNKH (SEQ ID NO: 26), NVPPIFNDVYWIA (SEQ ID NO: 27), RFRGLISLSQVYL (SEQ ID NO: 28), TKQSFDLSVKAQYKKN (SEQ ID NO: 29), VPPIFNDVYWI (SEQ ID NO: 30), FRGLISLSQVY (SEQ ID NO: 31), TKQSFDLSVKAQYKKN (SEQ ID NO: 32), PPIFNDVYW (SEQ ID NO: 33), RGLISLSQV (SEQ ID NO: 34), KQSFDLSVKAQYKK (SEQ ID NO: 35), RFRGLISLSQVYLDP (SEQ ID NO: 221), и SVCGCPVGHHDVVGL (SEQ ID NO: 222);
[00734] эпитоп, который включен в любую из последовательностей SEQ ID NO: 6 - SEQ ID NO: 35 и SEQ ID NO: 221 - SEQ ID NO: 222; или
[00735] последовательность, которая соответствует 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88% или более, 89% или более, 90% или более, 91% или более, 92% или более, 93% или более, 94% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более, или 100% или более любой из последовательностей, представленных SEQ ID NO: 6 - SEQ ID NO: 35 и от SEQ ID NO: 221 - SEQ ID NO: 222;
[00736] в которой а обозначает D-форму аланина, а Z обозначает 6-аминогексановую кислоту;
[00737] Т1 и Т2, каждый независимо выбирают из группы, состоящей из следующего:
[00738] последовательности, выбранной из группы, состоящей из K(Cha)VAAWTLKAA (SEQ ID NO: 1), PKYVKQNTLKLAT (SEQ ID NO: 2), ILMQYIKANSKFIGI (SEQ ID NO: 3), QSIALSSLMVAQAIP (SEQ ID NO: 4), ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 5), PLGFFPDHQL (SEQ ID NO: 162), WPEANQVGAGAFGPGF (SEQ ID NO: 163), MQWNSTALHQALQDP (SEQ ID NO: 164), MQWNSTTFHQTLQDPRVRGLYFPAGG (SEQ ID NO: 165), FFLLTRILTI (SEQ ID NO: 166), FFLLTRILTIPQSLD (SEQ ID NO: 167), TSLNFLGGTTVCLGQ (SEQ ID NO: 168), QSPTSNHSPTSCPPIC (SEQ ID NO: 169), IIFLFILLLCLIFLLVLLD (SEQ ID NO: 170), CTTPAQGNSMFPSC (SEQ ID NO: 171), CTKPTDGN (SEQ ID NO: 172), WASVRFSW (SEQ ID NO: 173), LLPIFFCLW (SEQ ID NO: 174), MDIDPYKEFGATVELLSFLP (SEQ ID NO: 175), FLPSDFFPSV (SEQ ID NO: 176), RDLLDTASALYREALESPEH (SEQ ID NO: 177), PHHTALRQAILCWGELMTLA (SEQ ID NO: 178), GRETVIEYLVSFGVW (SEQ ID NO: 179), EYLVSFGVWIRTPPA (SEQ ID NO: 180), VSFGVWIRTPPAYRPPNAPI (SEQ ID NO: 181), TVVRRRGRSP (SEQ ID NO: 182), VGPLTVNEKRRLKLI (SEQ ID NO: 183), RHYLHTLWKAGILYK (SEQ ID NO: 184), ESRLVVDFSQFSRGN (SEQ ID NO: 185), LQSLTNLLSSNLSWL (SEQ ID NO: 186), SSNLSWLSLDVSAAF (SEQ ID NO: 187), LHLYSHPIILGFRKI (SEQ ID NO: 188), KQCFRKLPVNRPIDW (SEQ ID NO: 189), LCQVFADATPTGWGL (SEQ ID NO: 190), AANWILRGTSFVYVP (SEQ ID NO: 191), EIRLKVFVLGGCRHK (SEQ ID NO: 192), KFVAAWTLKAA (SEQ ID NO: 195), KYVAAWTLKAA (SEQ ID NO: 196), DIEKKIAKMEKASSVFNVVNS (SEQ ID NO: 223), YSGPLKAEIAQRLEDV (SEQ ID NO: 224), K(Cha)VKANTLKAA (SEQ ID NO: 225), K(Cha)VKANTLKAA (SEQ ID NO: 226), K(Cha)VKAWTLKAA (SEQ ID NO: 227), K(Cha)VKAWTLKAA (SEQ ID NO: 228), K(Cha)VWANTLKAA (SEQ ID NO: 229), K(Cha)VWANTLKAA (SEQ ID NO: 230), K(Cha)VWAYTLKAA (SEQ ID NO: 231), K(Cha)VWAVTLKAA (SEQ ID NO: 232), K(Cha)VYAWTLKAA (SEQ ID NO: 233), K(Cha)VYAWTLKAA (SEQ ID NO: 234), R(Cha)VRANTLKAA (SEQ ID NO: 235), K(Cha)VKAHTLKAA (SEQ ID NO: 236), K(Cha)VKAHTLKAA (SEQ ID NO: 237), K(Cha)VAANTLKAA (SEQ ID NO: 238), K(Cha)VAANTLKAA (SEQ ID NO: 239), K(Cha)VAAYTLKAA (SEQ ID NO: 240), K(Cha)VAAYTLKAA (SEQ ID NO: 241), K(Cha)VAAWTLKAA (SEQ ID NO: 242), K(Cha)VAAKTLKAA (SEQ ID NO: 243), K(Cha)VAAHTLKAA (SEQ ID NO: 244), K(Cha)VAAATLKAA (SEQ ID NO: 245), K(Cha)VAAWTLKAA (SEQ ID NO: 246), и K(Cha)VMAATLKAA (SEQ ID NO: 247);
[00739] последовательности, представленной следующей [Формулой I] или [Формулой II]:
[00740] [Формула I] N-Lys-X1-X2-Ala-Ala-X3-Thr-X4-X5-Ala-Ala-C
[00741] в которой X1 представляет собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин;
[00742] X2 представляет собой гидрофобную аминокислоту, или лейцин (Leu), или изолейцин (Ile);
[00743] X3 представляет собой ароматическую или циклическую аминокислоту или фенилаланин (Phe), тирозин (Tyr) или гистидин (His);
[00744] X4 представляет собой алифатическую аминокислоту с длинной цепью, или изолейцин (Ile), или валин (Val); и
[00745] X5 представляет собой заряженную аминокислоту или аргинин (Arg), лейцин (Leu), аспарагиновую кислоту (Asp), глутамин (Gln) или глицин (Gly);
[00746] [Формула II] N-Lys-X1-Val-X2-Ala-X3-Thr-Leu-Lys-Ala-Ala-C:
[00747] в которой X1 представляет собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин;
[00748] X2 представляет собой лизин (Lys), триптофан (Trp), тирозин (Tyr), аргинин (Arg), аланин (Ala) или метионин (Met);
[00749] X3 представляет собой аспарагин (Asn), триптофан (Trp), тирозин (Tyr), валин (Val), гистидин (His), лизин (Lys) или аланин (Ala); и
[00750] последовательности, которая соответствует 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88% или более, 89% или более, 90% или более, 91% или более, 92% или более, 93% или более, 94% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более, или 100% или более любой из последовательностей, представленных SEQ ID NO: 1 - SEQ ID NO: 5, SEQ ID NO: 162 - SEQ ID NO: 192, SEQ ID NO: 195 - SEQ ID NO: 196, SEQ ID NO: 223 - SEQ ID NO: 247 и [формулой I] или [формулой II];
[00751] в которых (Cha) обозначает L-циклогексилаланины, а X обозначает любую стандартную аминокислоту.
[00752] Пример 89. Высшая концепция формулы Единицы-E
[00753] Пептидная единица любого из примеров 1 и 2, где пептидная единица представлена следующей[формулой E]:
[00754][Формула E] A1-B1-A2-T1-A3-T2-A4-B2-A5
[00755] в которой пептидная единица представляет собой единицу, которая может индуцировать гуморальный иммунитет путем распознавания CD4+Т-клетками и имеет длину, соответствующую 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру, 32-меру, 33-меру, 34-меру, 35-меру, 36-меру, 37-меру, 38-меру, 39-меру, 40-меру, 41-меру, 42-меру, 43-меру, 44-меру, 45-меру, 46-меру, 47-меру, 48-меру, 49-меру, 50-меру, 51-меру, 52-меру, 53-меру, 54-меру, 55-меру, 56-меру, 57-меру, 58-меру, 59-меру, 60-меру, 61-меру, 62-меру, 63-меру, 64-меру, 65-меру, 66-меру, 67-меру, 68-меру, 69-меру, 70-меру или 71-меру;
[00756] в которых А1 представляет собой первую вспомогательную часть или отсутствует; где первая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00757] А2 представляет собой вторую вспомогательную часть или отсутствует; где вторая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и необязательно может содержать нестандартную аминокислоту;
[00758] А3 представляет собой третью вспомогательную часть или отсутствует; где третья вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00759] А4 представляет собой четвертую вспомогательную часть или отсутствует; где четвертая вспомогательная часть имеет линкерную функцию, защитную функцию, функцию образования циклической формы, фиктивную функцию и/или функцию повышения растворимости и может необязательно содержать нестандартную аминокислоту;
[00760] В1, который представляет собой В-клеточный эпитоп, представляет собой фрагмент аполипопротеина В-100 или мимотоп аполипопротеина В-100 и может индуцировать антитела, нацеленные на аполипопротеин В-100;
[00761] В2, который представляет собой В-клеточный эпитоп, представляет собой фрагмент аполипопротеина В-100 или мимотоп аполипопротеина В-100 и может индуцировать антитела, нацеленные на аполипопротеин В-100;
[00762] Т1, который представляет собой Th-эпитоп, отличается тем, что он может распознаваться CD4+Т-клетками и имеет длину, соответствующую 8-меру, 9-меру, 10-меру, 11-меру, 12-меру, 13-меру, 14-меру, 15-меру, 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру или 32-меру; и
[00763] Т2, который представляет собой Th-эпитоп, отличается тем, что он может распознаваться CD4+Т-клетками и имеет длину, соответствующую 8-меру, 9-меру, 10-меру, 11-меру, 12-меру, 13-меру, 14-меру, 15-меру, 16-меру, 17-меру, 18-меру, 19-меру, 20-меру, 21-меру, 22-меру, 23-меру, 24-меру, 25-меру, 26-меру, 27-меру, 28-меру, 29-меру, 30-меру, 31-меру или 32-меру; и
[00764] Пример 90. Комбинация последовательностей Единицы-E
[00765] В примере 89
[00766] A1, A2, A3 и A4, каждый независимо выбран из группы, состоящей из a, Z, aZ, Za, RN, AF, CR, LS, KT, KH, RF, DP, SV, GL, ZRNV (SEQ ID NO: 36), aZRN (SEQ ID NO: 37), IAFZ (SEQ ID NO: 38), AFZa (SEQ ID NO: 39), RNVP (SEQ ID NO: 40), WIAF (SEQ ID NO: 41), ZCRF (SEQ ID NO: 42), aZCR (SEQ ID NO: 43), YLSZ (SEQ ID NO: 44), LSZa (SEQ ID NO: 45), CRFR (SEQ ID NO: 46), VYLS (SEQ ID NO: 47), ZKTT (SEQ ID NO: 48), aZKT (SEQ ID NO: 49), NKHZ (SEQ ID NO: 50), KHZa (SEQ ID NO: 51), GSHHHHHHGSDDDDK (SEQ ID NO: 52), HHHHHH (SEQ ID NO: 53), MRGSHHHHHHGSDDDDKIVD (SEQ ID NO: 54), GGGGSGGGGGGSS (SEQ ID NO: 55), RRRRRR (SEQ ID NO: 159), GSHHHHHHGSDDDDKZa (SEQ ID NO: 193), и aZGSHHHHHHGSDDDDK (SEQ ID NO: 194), либо отсутствует; в которых а обозначает D-форму аланина, а Z обозначает 6-аминогексановую кислоту;
[00767] B1 и B2, каждый независимо, содержат последовательность, выбранную из группы, состоящей из;
[00768] последовательности, выбранной из группы, состоящей из RNVPPIFNDVYWIAF (SEQ ID NO: 6), CRFRGLISLSQVYLS (SEQ ID NO: 7), KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8), RNVPPIFNDVY (SEQ ID NO: 9), CRFRGLISLSQ (SEQ ID NO: 10), KTTKQSFDLSVK (SEQ ID NO: 11), RNVPPIFNDVYW (SEQ ID NO: 12), CRFRGLISLSQV (SEQ ID NO: 13), KTTKQSFDLSVKAQYKK (SEQ ID NO: 14), RNVPPIFNDVYWI (SEQ ID NO: 15), CRFRGLISLSQVY (SEQ ID NO: 16), KTTKQSFDLSVKAQYKKN (SEQ ID NO: 17), PIFNDVYWIAF (SEQ ID NO: 18), GLISLSQVYLS (SEQ ID NO: 19), QSFDLSVKAQYKKNKH (SEQ ID NO: 20), PPIFNDVYWIAF (SEQ ID NO: 21), RGLISLSQVYLS (SEQ ID NO: 22), KQSFDLSVKAQYKKNKH (SEQ ID NO: 23), VPPIFNDVYWIAF (SEQ ID NO: 24), FRGLISLSQVYLS (SEQ ID NO: 25), TKQSFDLSVKAQYKKNKH (SEQ ID NO: 26), NVPPIFNDVYWIA (SEQ ID NO: 27), RFRGLISLSQVYL (SEQ ID NO: 28), TKQSFDLSVKAQYKKN (SEQ ID NO: 29), VPPIFNDVYWI (SEQ ID NO: 30), FRGLISLSQVY (SEQ ID NO: 31), TKQSFDLSVKAQYKKN (SEQ ID NO: 32), PPIFNDVYW (SEQ ID NO: 33), RGLISLSQV (SEQ ID NO: 34), KQSFDLSVKAQYKK (SEQ ID NO: 35), RFRGLISLSQVYLDP (SEQ ID NO: 221), и SVCGCPVGHHDVVGL (SEQ ID NO: 222);
[00769] эпитоп, который включен в любую из последовательностей SEQ ID NO: 6 - SEQ ID NO: 35 и SEQ ID NO: 221 - SEQ ID NO: 222; или
[00770] последовательность, которая соответствует 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88% или более, 89% или более, 90% или более, 91% или более, 92% или более, 93% или более, 94% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более, или 100% или более любой из последовательностей, представленных SEQ ID NO: 6 - SEQ ID NO: 35 и от SEQ ID NO: 221 - SEQ ID NO: 222;
[00771] в которых а обозначает D-форму аланина, а Z обозначает 6-аминогексановую кислоту;
[00772] Т1 и Т2, каждый независимо выбирают из группы, состоящей из следующего:
[00773] последовательности, выбранной из группы, состоящей из K(Cha)VAAWTLKAA (SEQ ID NO: 1), PKYVKQNTLKLAT (SEQ ID NO: 2), ILMQYIKANSKFIGI (SEQ ID NO: 3), QSIALSSLMVAQAIP (SEQ ID NO: 4), ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ (SEQ ID NO: 5), PLGFFPDHQL (SEQ ID NO: 162), WPEANQVGAGAFGPGF (SEQ ID NO: 163), MQWNSTALHQALQDP (SEQ ID NO: 164), MQWNSTTFHQTLQDPRVRGLYFPAGG (SEQ ID NO: 165), FFLLTRILTI (SEQ ID NO: 166), FFLLTRILTIPQSLD (SEQ ID NO: 167), TSLNFLGGTTVCLGQ (SEQ ID NO: 168), QSPTSNHSPTSCPPIC (SEQ ID NO: 169), IIFLFILLLCLIFLLVLLD (SEQ ID NO: 170), CTTPAQGNSMFPSC (SEQ ID NO: 171), CTKPTDGN (SEQ ID NO: 172), WASVRFSW (SEQ ID NO: 173), LLPIFFCLW (SEQ ID NO: 174), MDIDPYKEFGATVELLSFLP (SEQ ID NO: 175), FLPSDFFPSV (SEQ ID NO: 176), RDLLDTASALYREALESPEH (SEQ ID NO: 177), PHHTALRQAILCWGELMTLA (SEQ ID NO: 178), GRETVIEYLVSFGVW (SEQ ID NO: 179), EYLVSFGVWIRTPPA (SEQ ID NO: 180), VSFGVWIRTPPAYRPPNAPI (SEQ ID NO: 181), TVVRRRGRSP (SEQ ID NO: 182), VGPLTVNEKRRLKLI (SEQ ID NO: 183), RHYLHTLWKAGILYK (SEQ ID NO: 184), ESRLVVDFSQFSRGN (SEQ ID NO: 185), LQSLTNLLSSNLSWL (SEQ ID NO: 186), SSNLSWLSLDVSAAF (SEQ ID NO: 187), LHLYSHPIILGFRKI (SEQ ID NO: 188), KQCFRKLPVNRPIDW (SEQ ID NO: 189), LCQVFADATPTGWGL (SEQ ID NO: 190), AANWILRGTSFVYVP (SEQ ID NO: 191), EIRLKVFVLGGCRHK (SEQ ID NO: 192), KFVAAWTLKAA (SEQ ID NO: 195), KYVAAWTLKAA (SEQ ID NO: 196), DIEKKIAKMEKASSVFNVVNS (SEQ ID NO: 223), YSGPLKAEIAQRLEDV (SEQ ID NO: 224), K(Cha)VKANTLKAA (SEQ ID NO: 225), K(Cha)VKANTLKAA (SEQ ID NO: 226), K(Cha)VKAWTLKAA (SEQ ID NO: 227), K(Cha)VKAWTLKAA (SEQ ID NO: 228), K(Cha)VWANTLKAA (SEQ ID NO: 229), K(Cha)VWANTLKAA (SEQ ID NO: 230), K(Cha)VWAYTLKAA (SEQ ID NO: 231), K(Cha)VWAVTLKAA (SEQ ID NO: 232), K(Cha)VYAWTLKAA (SEQ ID NO: 233), K(Cha)VYAWTLKAA (SEQ ID NO: 234), R(Cha)VRANTLKAA (SEQ ID NO: 235), K(Cha)VKAHTLKAA (SEQ ID NO: 236), K(Cha)VKAHTLKAA (SEQ ID NO: 237), K(Cha)VAANTLKAA (SEQ ID NO: 238), K(Cha)VAANTLKAA (SEQ ID NO: 239), K(Cha)VAAYTLKAA (SEQ ID NO: 240), K(Cha)VAAYTLKAA (SEQ ID NO: 241), K(Cha)VAAWTLKAA (SEQ ID NO: 242), K(Cha)VAAKTLKAA (SEQ ID NO: 243), K(Cha)VAAHTLKAA (SEQ ID NO: 244), K(Cha)VAAATLKAA (SEQ ID NO: 245), K(Cha)VAAWTLKAA (SEQ ID NO: 246), и K(Cha)VMAATLKAA (SEQ ID NO: 247);
[00774] последовательности, представленной следующей [Формулой I] или [Формулой II]:
[00775] [Формула I] N-Lys-X1-X2-Ala-Ala-X3-Thr-X4-X5-Ala-Ala-C
[00776] в которой X1 представляет собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин;
[00777] X2 представляет собой гидрофобную аминокислоту, или лейцин (Leu), или изолейцин (Ile);
[00778] X3 представляет собой ароматическую или циклическую аминокислоту или фенилаланин (Phe), тирозин (Tyr) или гистидин (His);
[00779] X4 представляет собой алифатическую аминокислоту с длинной цепью, или изолейцин (Ile), или валин (Val); и
[00780] X5 представляет собой заряженную аминокислоту или аргинин (Arg), лейцин (Leu), аспарагиновую кислоту (Asp), глутамин (Gln) или глицин (Gly);
[00781] [Формула II] N-Lys-X1-Val-X2-Ala-X3-Thr-Leu-Lys-Ala-Ala-C:
[00782] в которой X1 представляет собой тирозин (Tyr), фенилаланин (Phe) или L-циклогексилаланин;
[00783] X2 представляет собой лизин (Lys), триптофан (Trp), тирозин (Tyr), аргинин (Arg), аланин (Ala) или метионин (Met); и
[00784] X3 представляет собой аспарагин (Asn), триптофан (Trp), тирозин (Tyr), валин (Val), гистидин (His), лизин (Lys) или аланин (Ala); и
[00785] последовательности, которая соответствует 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88% или более, 89% или более, 90% или более, 91% или более, 92% или более, 93% или более, 94% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более, или 100% или более любой из последовательностей, представленных SEQ ID NO: 1 - SEQ ID NO: 5, SEQ ID NO: 162 - SEQ ID NO: 192, SEQ ID NO: 195 - SEQ ID NO: 196, SEQ ID NO: 223 - SEQ ID NO: 247 и [формулой I] или [формулой II];
[00786] в которых (Cha) обозначает L-циклогексилаланины, а X обозначает любую стандартную аминокислоту.
[00787] пептид, включающий пептидную единицу
[00788] Пример 91, пептид, включающий 2 или более пептидных единиц
[00789] Пептид, в котором связаны 2 или более пептидных единиц любого из примеров 1-90.
[00790] Пример 92, пептид, включающий от двух до восьми пептидных единиц.
[00791] Пептид, в котором связаны 2, 3, 4, 5, 6, 7 или 8 пептидных единиц любого из примеров от 1 до 90.
[00792] Пример 93. Последовательность конкатемера
[00793] Пептид из Примера 91, где каждая из пептидных единиц имеет одинаковую или эквивалентную последовательность.
[00794] Пример 94. Нитка бус
[00795] Пептид из примера 91, где каждая из пептидных единиц имеет отличную последовательность.
[00796] Пример 95. Циклическая форма
[00797] Пептид Примера 91, в котором пептид отличается тем, что он дополнительно включает вспомогательную часть, обладающую функцией образования циклической формы на N-конце и С-конце, и пептид образует циклическую форму посредством вспомогательной части.
[00798] нуклеиновая кислота, кодирующая пептидную единицу и/или пептид
[00799] Пример 96. Кодирующая нуклеиновая кислота, не включающая нестандартную аминокислоту
[00800] Нуклеиновая кислота, которая кодирует пептидную единицу любого из примеров 1-6 и/или пептид любого из примеров 91-95, в которых пептидная единица и пептид не включают нестандартную аминокислоту.
[00801] Пример 97. Кодирующая нуклеиновая кислота формулы каждой единицы, не включающая нестандартную аминокислоту
[00802] Нуклеиновая кислота, которая кодирует пептидную единицу любого из примеров от примера 7 до примера 10, от примера 14 до примера 18, примера 22, от примера 26 до примера 30, примера 34, от примера 38 до примера 41, примера 45, от примера 49 до примера 54, от примера 58 до примера 60, примера 64, от примера 68 до примера 72, в которых пептидная единица отличается тем, что она не включает нестандартную аминокислоту.
[00803] Пример 98. Ограничение по последовательности пептидной единицы.
[00804] Нуклеиновая кислота любого из примеров 96 и 97, где нуклеиновая кислота кодирует пептидную единицу, выбранную из следующего: RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXCRFRGLISLSQVYLS (SEQ ID NO: 198); RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXGSHHHHHHGSDDDDK (SEQ ID NO: 199); GSHHHHHHGSDDDDKXXKXVAAWTLKAAXXRNVPPIFNDVYWIAF (SEQ ID NO: 200); KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXCRFRGLISLSQVYLS (SEQ ID NO: 201); RNVPPIFNDVYWIAFCRFRGLISLSQVYLSXXKXVAAWTLKAAXX (SEQ ID NO: 202); RNVPPIFNDVYWIAFXPKYVKQNTLKLATXCRFRGLISLSQVYLS (SEQ ID NO: 203); RNVPPIFNDVYWIAFXXKXVAAWTLKAAXX (SEQ ID NO: 204); RNVPPIFNDVYWIAFKXVAAWTLKAA (SEQ ID NO: 205); RNVPPIFNDVYWIAFKXVAAWTLKAAHHHHHH (SEQ ID NO: 206); RNVPPIFNDVYWIAFXXKXVAAWTLKAACRFRGLISLSQVYLS (SEQ ID NO: 207); RNVPPIFNDVYWIAFXXKXVAAWTLKAACR (SEQ ID NO: 208); RNVPPIFNDVYWIAFXXKFVAAWTLKAAXXCRFRGLISLSQVYLS (SEQ ID NO: 209); RNVPPIFNDVYWIAFXXKFVAAWTLKAAXX (SEQ ID NO: 210); RNVPPIFNDVYWIAFXXKFVAAWTLKAACRFRGLISLSQVYLS (SEQ ID NO: 211); и RNVPPIFNDVYWIAFXXKFVAAWTLKAACR (SEQ ID NO: 212), в которых X обозначает любую стандартную аминокислоту.
[00805] Пример 99. Ограничение по последовательности ДНК, кодирующей единицу
[00806] Нуклеиновая кислота Примера 98 представляет собой ДНК, которая представлена последовательностью, выбранной из следующего:
[00807] 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 248); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 249); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 250); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNGGATCGCATCACCATCACCATCACGGATCCGATGATGATGACAAG-3' (SEQ ID NO: 251); 5'-ACGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNGGATCGCATCACCATCACCATCACGGATCCGATGATGATGACAAG-3' (SEQ ID NO: 252); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNGGATCGCATCACCATCACCATCACGGATCCGATGATGATGACAAG-3' (SEQ ID NO: 253); 5'-GGATCGCATCACCATCACCATCACGGATCCGATGATGATGACAAGNNNNNNAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNCGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTC-3' (SEQ ID NO: 254); 5'-GGATCGCATCACCATCACCATCACGGATCCGATGATGATGACAAGNNNNNNAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNCGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTC-3' (SEQ ID NO: 255); 5'-GGATCGCATCACCATCACCATCACGGATCCGATGATGATGACAAGNNNNNNAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNCGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTC-3' (SEQ ID NO: 256); 5'-AAAACGACAAAGCAATCATTTGATTTAAGTGTAAAAGCTCAGTATNNNNNNAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 257); 5'-AAAACGACAAAGCAATCATTTGATTTAAGTGTAAAAGCTCAGTATNNNNNNAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 258); 5'-AAAACGACAAAGCAATCATTTGATTTAAGTGTAAAAGCTCAGTATNNNNNNAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNNTGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 259); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCTGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCCNNNNNNAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 260); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCTGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCCNNNNNNAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 261); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCTGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCCNNNNNNAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 262); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNCCTAAGTATGTGAAGCAGAATACACTGAAGCTGGCAACCNNNTGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 263); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 264); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 265); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCANNNNNN-3' (SEQ ID NO: 266); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCA-3' (SEQ ID NO: 267); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCA-3' (SEQ ID NO: 268); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCA-3' (SEQ ID NO: 269); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCACATCACCATCACCATCAC-3' (SEQ ID NO: 270); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCACATCACCATCACCATCAC-3' (SEQ ID NO: 271); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCACATCACCATCACCATCAC-3' (SEQ ID NO: 272); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 273); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 274); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGTTTCCGTGGACTGATTTCCCTGTCCCAGGTTTATCTGTCC-3' (SEQ ID NO: 275); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGNNNGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGT-3' (SEQ ID NO: 276); 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGTTCGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGT-3' (SEQ ID NO: 277); и 5'-CGTAATGTTCCTCCTATCTTCAATGATGTTTATTGGATTGCATTCNNNNNNAAGTATGTGGCAGCTTGGACCCTGAAGGCAGCATGCCGT-3' (SEQ ID NO: 278).
[00808] Пример 100. Ограничение по последовательности РНК, кодирующей единицу
[00809] Нуклеиновая кислота примера 98, РНК, представленная последовательностью, выбранной из следующего:
[00810] 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGNNNGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCC-3' (SEQ ID NO: 279); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGUUCGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCC-3' (SEQ ID NO: 280); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGUAUGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCC-3' (SEQ ID NO: 281); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGNNNGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNGGAUCGCAUCACCAUCACCAUCACGGAUCCGAUGAUGAUGACAAG-3' (SEQ ID NO: 282); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGUUCGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNGGAUCGCAUCACCAUCACCAUCACGGAUCCGAUGAUGAUGACAAG-3' (SEQ ID NO: 283); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGUAUGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNGGAUCGCAUCACCAUCACCAUCACGGAUCCGAUGAUGAUGACAAG-3' (SEQ ID NO: 284); 5'-GGAUCGCAUCACCAUCACCAUCACGGAUCCGAUGAUGAUGACAAGNNNNNNAAGNNNGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNCGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUC-3' (SEQ ID NO: 285); 5'-GGAUCGCAUCACCAUCACCAUCACGGAUCCGAUGAUGAUGACAAGNNNNNNAAGUUCGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNCGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUC-3' (SEQ ID NO: 286); 5'-GGAUCGCAUCACCAUCACCAUCACGGAUCCGAUGAUGAUGACAAGNNNNNNAAGUAUGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNCGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUC-3' (SEQ ID NO: 287); 5'-AAAACGACAAAGCAAUCAUUUGAUUUAAGUGUAAAAGCUCAGUAUNNNNNNAAGNNNGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCC-3' (SEQ ID NO: 288); 5'-AAAACGACAAAGCAAUCAUUUGAUUUAAGUGUAAAAGCUCAGUAUNNNNNNAAGUUCGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCC-3' (SEQ ID NO: 289); 5'-AAAACGACAAAGCAAUCAUUUGAUUUAAGUGUAAAAGCUCAGUAUNNNNNNAAGUAUGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNNUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCC-3' (SEQ ID NO: 290); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCCNNNNNNAAGNNNGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNN-3' (SEQ ID NO: 291); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCCNNNNNNAAGUUCGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNN-3' (SEQ ID NO: 292); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCCNNNNNNAAGUAUGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNN-3' (SEQ ID NO: 293); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNCCUAAGUAUGUGAAGCAGAAUACACUGAAGCUGGCAACCNNNUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCC-3' (SEQ ID NO: 294); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGNNNGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNN-3' (SEQ ID NO: 295); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGUUCGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNN-3' (SEQ ID NO: 296); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGUAUGUGGCAGCUUGGACCCUGAAGGCAGCANNNNNN-3' (SEQ ID NO: 297); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCAAGNNNGUGGCAGCUUGGACCCUGAAGGCAGCA-3' (SEQ ID NO: 298); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCAAGUUCGUGGCAGCUUGGACCCUGAAGGCAGCA-3' (SEQ ID NO: 299); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCAAGUAUGUGGCAGCUUGGACCCUGAAGGCAGCA-3' (SEQ ID NO: 300); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCAAGNNNGUGGCAGCUUGGACCCUGAAGGCAGCACAUCACCAUCACCAUCAC-3' (SEQ ID NO: 301); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCAAGUUCGUGGCAGCUUGGACCCUGAAGGCAGCACAUCACCAUCACCAUCAC-3' (SEQ ID NO: 302); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCAAGUAUGUGGCAGCUUGGACCCUGAAGGCAGCACAUCACCAUCACCAUCAC-3' (SEQ ID NO: 303); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGNNNGUGGCAGCUUGGACCCUGAAGGCAGCAUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCC-3' (SEQ ID NO: 304); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGUUCGUGGCAGCUUGGACCCUGAAGGCAGCAUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCC-3' (SEQ ID NO: 305); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGUAUGUGGCAGCUUGGACCCUGAAGGCAGCAUGCCGUUUCCGUGGACUGAUUUCCCUGUCCCAGGUUUAUCUGUCC-3' (SEQ ID NO: 306); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGNNNGUGGCAGCUUGGACCCUGAAGGCAGCAUGCCGU-3' (SEQ ID NO: 307); 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGUUCGUGGCAGCUUGGACCCUGAAGGCAGCAUGCCGU-3' (SEQ ID NO: 308); и 5'-CGUAAUGUUCCUCCUAUCUUCAAUGAUGUUUAUUGGAUUGCAUUCNNNNNNAAGUAUGUGGCAGCUUGGACCCUGAAGGCAGCAUGCCGU-3' (SEQ ID NO: 309).
[00811] Пример 101, Вектор
[00812] Вектор, включающий любую нуклеиновую кислоту из примеров 96-100.
[00813] Пример 102, Ограничение по Вектору
[00814] Вектор Примера 101, где вектор выбран из группы, состоящей из плазмид, ретровирусов, лентивирусов, аденовирусов, аденоассоциированных вирусов, вирусов коровьей оспы, поксвирусов и вирусов простого герпеса.
[00815] Пример 103. Кодон-оптимизированная нуклеиновая кислота млекопитающих
[00816] Нуклеиновая кислота любого из Примеров 96-98, где нуклеиновая кислота оптимизирована по кодонам для видов, выбранных из млекопитающих.
[00817] Пример 104. Кодон-оптимизированная нуклеиновая кислота человека
[00818] Нуклеиновая кислота любого из примеров 96-98, где нуклеиновая кислота оптимизирована по кодонам человека.
[00819] Пример 105, Прокариотическая кодон-оптимизированная нуклеиновая кислота
[00820] Нуклеиновая кислота любого из примеров 96-98, где нуклеиновая кислота оптимизирована по кодонам для видов, выбранных из прокариот.
[00821] Пример 106. Кодон-оптимизированная нуклеиновая кислота E.coli.
[00822] Нуклеиновая кислота любого из Примеров 96-98, отличающаяся тем, что нуклеиновая кислота оптимизирована по кодонам E.coli.
[00823] фармацевтическая композиция, включающая пептид
[00824] Пример 107. Фармацевтическая композиция для иммунотерапии.
[00825] Фармацевтическая композиция для иммунотерапии, включающая следующее:
[00826] пептидную единицу любого из Примеров 1-90 и/или пептид любого из Примеров 91-95; и
[00827] адъюванты.
[00828] Пример 108. Фармацевтическая композиция для лечения ожирения
[00829] Фармацевтическая композиция для лечения ожирения, включающая следующее:
[00830] пептидную единицу любого из Примеров 1-90 и/или пептид любого из Примеров 91-95; и
[00831] адъюванты.
[00832] Пример 109. Ограничение по адъювантам
[00833] В любом из примеров 107-108 адъювант представляет собой воду, физиологический раствор, декстрозу, этанол, глицерин, хлорид натрия, декстрозу, маннит, сорбит, лактозу, желатин, альбумин, гидроксид алюминия, неполный адъювант Фрейнда и полный адъювант (Pifco Laboratories, Детройт, штат Мичиган), антигенный адъювант Merck 65 (Merck and Company, Inc., Рэуэй, штат Нью-Джерси), альгидрогель (Al(OH)3), гель гидроксида алюминия (квасцы) или соли алюминия, такие как фосфат алюминия, AS04 серия, MF, сквален, MF59, QS21, соли кальция, железа или цинка, нерастворимые суспензии ацилированного тирозина, ацилированной фруктозы, полисахариды катионного или анионного происхождения, полифосфазены, биоразлагаемые микросферы, Quil A, агонисты toll-подобных рецепторов (TLR), PHAD[Avanti полярный липид, монофосфорил липид A (синтетический)], монофосфорил липид A (MPL, монофосфорил липид A), синтетический липид A, имитаторы или аналоги липида A, соли алюминия, цитокины, сапонины, пролактин, гормон роста дезоксихолевая кислота, бетаглюкан, полирибонуклеотиды, производные мурамилдипептида (MDP), олигонуклеотиды CpG, липополисахарид грамотрицательных бактерий (LPS), полифосфазен, эмульсии, виросомы, кохлеат, микрочастицы поли(лактид-со-гликолида) (PLG), частицы полоксамера, микрочастицы, липосомы или соответствующие комбинации из этого.
[00834] фармацевтическая композиция, включающая кодирующую нуклеиновую кислоту
[00835] Пример 110. Готовая к применению кодирующая нуклеиновая кислота
[00836] Готовая к применению кодирующая нуклеиновая кислота, отличающаяся тем, что нуклеиновая кислота любого из примеров 96-106 приготовлена с использованием вирусного вектора и/или невирусного вектора.
[00837] Пример 111, Ограничение по вирусному вектору
[00838] В Примере 110 кодирующая нуклеиновая кислота отличается тем, что вирусный вектор выбран из следующего:
[00839] ретровируса; лентивируса; аденовируса; аденоассоциированного вируса; вируса коровьей оспы; поксвируса; и вируса простого герпеса.
[00840] Пример 112. Ограничение по формам готовой к применению нуклеиновой кислоты
[00841] В примере 110 кодирующая нуклеиновая кислота отличается тем, что готовая к применению нуклеиновая кислота выбрана из следующего:
[00842] депротеинизированной нуклеиновой кислоты; катионного пептида в комплексе с нуклеиновой кислотой (протамин); положительно заряженной катионной наноэмульсии масло-вода, включающей нуклеиновую кислоту (катионная наноэмульсия); нуклеиновой кислоты, связанной с химически модифицированным дендримером и образующей комплекс с полиэтиленгликолем и ПЭГ-липидом (наночастица модифицированного дендримера); нуклеиновой кислоты в комплексе с протамином в ПЭГ-липидных наночастицах (протаминовая липосома); нуклеиновой кислоты, образующей комплекс с катионным полимером, таким как полиэтиленимин и PEI (катионный полимер); нуклеиновой кислоты, образующей комплекс с катионным полимером, таким как PEI, и липидный компонент (катионная полимерная липосома); нуклеиновой кислоты, образующей комплекс с полисахаридным полимером, таким как хитозан (полисахаридная частица); нуклеиновой кислоты, образующей комплекс с полимером катионных липидных наночастиц (катионные липидные наночастицы); нуклеиновой кислоты, образующей комплекс с катионным липидом и холестерином (катионная липидно-холестериновая наночастица); и нуклеиновой кислоты, образующей комплекс с катионным липидом, холестерином и ПЭГ-липидом (катионный липид-холестерин-ПЭГ-наночастица).
[00843] Пример 113. Фармацевтическая композиция для иммунотерапии, включающая готовую к применению нуклеиновую кислоту.
[00844] Фармацевтическая композиция для иммунотерапии, включающая следующее:
[00845] готовую к применению нуклеиновую кислоту любого из примеров 100-112; и
[00846] адъюванты.
[00847] Пример 114. Фармацевтическая композиция для лечения ожирения, включающая готовую к применению нуклеиновую кислоту.
[00848] Фармацевтическая композиция для лечения ожирения, включающая следующее:
[00849] готовую к применению нуклеиновую кислоту любого из примеров 100-112; и
[00850] адъюванты.
[00851] Пример 115. Ограничение по адъювантам
[00852] Фармацевтическая композиция любого из примеров 113 и 114, где адъювант представляет собой одно или более из следующего:
[00853] липидные наночастицы (LNP); соли алюминия; хлорид 1,2-диолеил-3-триметиламмонийпропана; адъювант MF59 (Novartis); CD70; лиганд CD40 (CD40L); TriMix; протамин, действующий через передачу сигналов TLR7; и/или монофосфориллипид A бактериального происхождения.
[00854] Пример 116, включающие дополнительные ингредиенты
[00855] Фармацевтическая композиция любого из примеров 113-114, где фармацевтическая композиция включает один или более дополнительных ингредиентов, выбранных из следующих:
[00856] липиды; соли для балансировки кислотности организма; сахароза для сохранения стабильности при многократном замораживании-оттаивании; и вещества, повышающие стабильность вакцин.
[00857] Пример 117. Ограничение на дополнительные ингредиенты
[00858] Фармацевтическая композиция Примера 116,
[00859] где фармацевтическая композиция отличается тем, что липид представляет собой один или более липидов, выбранных из SM-102, PEG2000-DMG, DPSC, холестерина и ALC-0315;
[00860] соль представляет собой одну или более солей, выбранных из ацетата натрия, хлорида калия, дигидрофосфата калия, хлорида натрия и гидрофосфат натрия дигидрата; и
[00861] вещество, повышающее стабильность вакцины, представляет собой одно или более веществ, выбранных из уксусной кислоты, стабилизатора кислоты (трометамина) и этанола.
[00862] применение пептидов
[00863] Пример 118. Применение для иммунотерапии (первое медицинское применение).
[00864] Применение для иммунотерапии пептидной единицы любого из примеров 1-90, пептида любого из примеров 91-95, нуклеиновой кислоты любого из примеров 96-106 и/или фармацевтической композиции любого из примеров 107-109 и примеров 113-117.
[00865] Пример 119. Применение для лечения ожирения (первое медицинское применение).
[00866] Применение для лечения ожирения пептидной единицы любого из Примеров 1-90, пептида любого из Примеров 91-95, нуклеиновой кислоты любого из Примеров 96-106 и/или фармацевтической композиции любого из Примеров 107-109 и Примеров 113-117.
[00867] Пример 120. Применение для приготовления иммунотерапевтических средств (второе медицинское применение).
[00868] Применение для приготовления иммунотерапевтических средств пептидной единицы любого из Примеров 1-90, пептида любого из Примеров 91-95, нуклеиновой кислоты любого из Примеров 96-106 и/или фармацевтической композиции любого из примеров 107-109 и примеров 113-117.
[00869] Пример 121. Применение для приготовления терапевтических средств для лечения ожирения (второе медицинское применение).
[00870] Применение для приготовления терапевтических средств для лечения ожирения пептидной единицы любого из Примеров 1-90, пептида любого из Примеров 91-95, нуклеиновой кислоты любого из Примеров 96-106 и/или фармацевтической композиции любого из примеров 107-109 и примеров 113-117.
[00871] методы лечения с использованием пептидов
[00872] Пример 122. Иммунотерапия с использованием пептидов.
[00873] иммунотерапия, включающая следующее:
[00874] введение пептидной единицы любого из Примеров 1-90, пептида любого из Примеров 91-95, нуклеиновой кислоты любого из Примеров 96-106 и/или фармацевтической композиции любого из примеров 107-109 и примеров 113-примеров 117 в тело пациента.
[00875] Пример 123, Методы лечения ожирения с использованием пептидов
[00876] Методы лечения ожирения с использованием следующего:
[00877] введение пептидной единицы любого из Примеров 1-90, пептида любого из Примеров 91-95, нуклеиновой кислоты любого из Примеров 96-106 и/или фармацевтической композиции любого из примеров 107-109 и примеров 113-примеров 117 в тело пациента.
[00878] последовательности, сходные с пептидными единицами и/или пептидами
[00879] Пример 124. Последовательности, сходные с пептидными единицами.
[00880] Пептидная единица, которая имеет последовательность, соответствующую 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88%. или более, 89% или более, 90% или более, 91% или более, 92% или более, 93% или более, 94% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более или 100% или более любой одной пептидной единице из примеров 1-90.
[00881] Пример 125. Последовательности, сходные с пептидами.
[00882] Пептид, который имеет последовательность, соответствующую на 80% или более, 81% или более, 82% или более, 83% или более, 84% или более, 85% или более, 86% или более, 87% или более, 88% или более, 89% или более, 90% или более, 91% или более, 92% или более, 93% или более, 94% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более или 100% или более любой одной пептидной единицей из примеров 91-95.
[00883]
Экспериментальные Примеры
[00884] Далее изобретение, представленное настоящим описанием, будет описано более подробно посредством экспериментальных примеров и примеров. Эти примеры предназначены только для иллюстрации содержания, раскрытого в настоящем описании, и специалистам в данной области техники будет очевидно, что объем содержания, раскрытого в настоящем описании, не следует толковать как ограниченный этими примерами.
[00885]
[00886] Экспериментальный пример 1, экспериментальный метод
[00887] Экспериментальный пример 1.1. Получение пептидов.
[00888] Этот пептид был получен по запросу компании по синтезу пептидов (Anygen, Корея, город Кванджу). Пептид по настоящему изобретению может быть синтезирован с использованием общеизвестных способов (например, жидкофазный синтез пептидов, твердофазный синтез пептидов, конвергентный синтез коротких пептидных фрагментов и т.д.), и способ синтеза не ограничен. Например, пептид ОТР3 по настоящему изобретению может быть синтезирован с использованием конвергентного синтеза коротких пептидных фрагментов, при котором сайты длинноцепочечного пептида, с которыми можно легко осуществить связывание, виртуально расщепляются, и на основе этого получают различные части, а затем комбинируются друг с другом для окончательного синтеза желаемого пептида. Вышеупомянутый конвергентный синтез имеет ограничения, заключающиеся в том, что в пептидной последовательности должны присутствовать определенные аминокислоты, и, таким образом, пептид также может быть эффективно синтезирован с помощью метода комбинаторного пептидного синтеза, где подходящим образом объединяют метод синтеза в фазе раствора и метод синтеза пептида в твердой фазе.
[00889] Экспериментальный пример 1.2, подтверждение полученного пептида 1 - анализ чистоты
[00890] Чистоту пептида, полученного в экспериментальном примере 1.1, измеряли путем проведения ВЭЖХ-анализа (Shimadzu HPLC LabSolutions) с использованием обращенно-фазовой колонки C-18 (аналитическая колонка SHIMADZU C18). Что касается условий анализа, образец отделяли и проявляли в водном растворе 0,05% трифторацетата (ТФУ) и 0,05% растворе ТФУ в ацетотриле при 60°C, а затем чистоту подтверждали измерением пика поглощения при длине волны 230 нм.
[00891] Экспериментальный пример 1.3, подтверждение полученного пептида 2 - анализ молекулярной массы
[00892] Молекулярную массу пептида, полученного в экспериментальном примере 1.1, анализировали с помощью масс-спектрометра (AXIMA Assurance, MALDI-TOF, Shimadzu).
[00893] Экспериментальный пример 1.4, подтверждение полученного пептида 2 - количественный анализ
[00894] Пептид, полученный в экспериментальном примере 1.1, количественно определяли путем измерения коэффициента поглощения УФ-излучения (спектрофотометр Ultrospec 3000 Pro UV/VIS, Pharmacia). В частности, количественный анализ проводили с использованием коэффициента поглощения при 280 нм.
[00895] Экспериментальный пример 1.5. Приготовление композиции для введения in vivo.
[00896] Композицию для введения in vivo готовили путем смешивания пептида, полученного в экспериментальном примере 1.1, альгидрогеля (Al(OH)3, производства InvivoGe, Inc.) и PHAD (производства Avanti). Конкретный процесс заключается в следующем.
[00897] (1) Приготовленный пептидный порошок растворяли в 100% диметилсульфоксиде (ДМСО) до получения концентрации 100 мг/л.
[00898] (2) PBS добавляли к раствору пептид-ДМСО (1) и перемешивали для получения пептида в концентрации 50 мг/мл.
[00899] (3) PHAD растворяли в 100% ДМСО до концентрации 10 мг/мл, а затем разбавляли дистиллированной водой до концентрации 1 мг/мл.
[00900] (4) К смеси (2) добавляли альгидрогелевый адъювант (Invivogen, США) и раствор PHAD. Концентрация смешанной композиции составляла 50 мкг для пептида, 10 мкг для PHAD и 10% (об./об.) для адъюванта Alhydrogel на 100 мкл, что является количеством для одной дозы.
[00901] (5) После тщательного перемешивания смеси (4) полученную смесь подвергали реакции в течение ночи при перемешивании с помощью шейкера в помещении с низкой температурой (4°С).
[00902] (6) Для промывки DSMO реагенты (5) центрифугировали при 1400 об/мин в течение 15 минут и удаляли супернатант, за исключением примерно 1 мл над осадком. После этого туда добавляли 10 мл PBS и перемешивали.
[00903] (7) Процесс промывки (6) повторили 3 раза.
[00904] (8) После окончательного процесса промывки туда добавляли PBS, чтобы отрегулировать конечную концентрацию композиции. В частности, концентрация пептида составляла 50 мкг/100 мкл (в частности, 30 мкг/100 мкл в примере 4), концентрация PHAD составляла 10 мкг/100 мкл, а концентрация адъюванта альгидрогеля составляла 10% (об/об).
[00905] (9) Количество пептида, адсорбированного на алюминиевом геле, измеряли в соответствии с методом, описанным в экспериментальном примере 2.3. В результате было подтверждено, что скорость адсорбции составляет 95% или более, и пептид использовали в эксперименте.
[00906] Экспериментальный пример 1.6. Подготовка испытуемого
[00907] Для проверки действия композиции для введения in vivo, приготовленной в экспериментальном примере 1.5, виды C57BL/6, Balbc и/или ICR использовали в качестве экспериментальных мышей (приобретенных у Central Lab Animal Inc.). Несмотря на некоторые различия между видами, приобретенным мышам в среднем было 7 недель, они акклиматизировались в течение одной недели и их использовали для экспериментов, когда им было 8 недель. Экспериментальных мышей выращивали в условиях постоянной температуры и влажности окружающей среды в диапазоне температур 23±1°C, относительной влажности 50±5% и условиях, контролируемых в режиме 12-часов света/12-часов темноты. Питьевая вода и еда предоставлялись по требованию. В случае обычной диеты (приобретенной у Central Lab Animal Inc.) она состояла из 20% белков, 70% углеводов и 10% жиров в расчете на общее количество калорий, тогда как в случае вызывающей ожирение диеты (приобретенной у Research diets), она состоял из диеты с высоким содержанием жиров, включая 20% белков, 20% углеводов и 60% жиров в расчете на общее количество калорий. Экспериментальные мыши были разделены на каждую группу, чтобы иметь разные диетические условия и условия введения композиции, и были проведены эксперименты с количеством особей, которые могли быть статистически обработаны для каждой экспериментальной группы. Подробные условия описаны в каждом конкретном экспериментальном примере.
[00908] Экспериментальный пример 1.7. Введение испытуемому композиции, содержащей пептид
[00909] Испытуемому, подготовленному в экспериментальном примере 1.6, вводили разные композиции для каждой экспериментальной группы. Все композиции для введения вводили методом внутримышечной инъекции, и после дезинфекции мышц обоих бедер каждой мыши спиртовым тампоном вводили по 50 мкл каждой из общего количества 100 мкл.
[00910] Экспериментальный пример 1.8. Подтверждение эффекта композиции для введения in vivo 1 - Измерение массы
[00911] Чтобы подтвердить эффект композиции, введенной в организм в соответствии с экспериментальным примером 1.7, в отношении потери массы у испытуемого, для каждой экспериментальной группы измеряли массу тела и массу органов у мышей. От момента прибытия до конца эксперимента для каждой экспериментальной группы мышей рассчитывали среднее значение после трех измерений в неделю для получения среднего значения массы за каждую неделю. После завершения эксперимента каждую мышь подвергали анестезии, проводили диссекцию органов, измеряли массу и получали среднее значение для каждой экспериментальной группы.
[00912] Экспериментальный пример 1.9. Подтверждение эффекта композиции для введения in vivo. 2. Подтверждение титра антител.
[00913] Чтобы определить, индуцирует ли композиция для введения in vivo, введенная в экспериментальном примере 1.7, антитело против В-клеточного эпитопа, выполняли способ подтверждения титра антител с использованием целевого антигена, заключающийся в следующем. В частности, целевой антиген представляет собой RNVPPIFNDVYWIAF (SEQ ID NO: 6) или ApoB100. Время подтверждения титра антител может варьироваться по мере необходимости, и это время было специально описано для каждого экспериментального примера.
[00914] 1. Процесс реакции покрытия антигеном
[00915] 1-1) Для каждой инъекции композиции для введения in vivo испытуемому собирали примерно 200 мкл крови из хвостовой вены испытуемого через неделю после инъекции.
[00916] 1-2) Поместив собранную кровь при 4°С на один час, проводили центрифугирование образца крови при 14000 об/мин в течение 10 минут для отделения сыворотки, которая является супернатантом.
[00917] 1-3) Разводили целевой антиген до концентрации 50 мкг/100 мкл в буфере для покрытия (0,05 М, бикарбонат, рН 9,6) и вносили антиген в 96-луночный планшет в количестве 50 мкг на лунку, и давали им прореагировать в течение ночи при 4°C, чтобы пептид покрыл стенку лунки.
[00918] 1-4) Планшет, покрытый целевым антигеном, промывали три раза, используя 300 мкл фосфатно-солевого буфера (PBS)-T (0,05% с Tween-20) на лунку.
[00919] 2. Процесс блокировки реакции
[00920] 2-1) Добавляли по 300 мкл 0,5% блокирующего раствора казеина на лунку планшета и оставляют для реакции на ночь при 4°C.
[00921] 2-2) Промывали планшет три раза по 300 мкл PBS-T на лунку.
[00922] 3. Процесс реакции первичного антитела
[00923] 3-1) В качестве реакции с первичными антителами разбавляли отделенную сыворотку до соответствующей концентрации, добавляли ее в количестве 100 мкл на лунку и дают прореагировать при 37°C в течение одного часа. В частности, после первоначальной инъекции сыворотку подвергали серийному разведению, разбавляя ее от 1/20 до 1/1000 в эксперименте и от 1/500 до 1/10 ООО в зависимости от испытуемого во время эксперимента после второй-третьей инъекции. В качестве положительного контроля использовали моноклональное антитело против пептида SEQ ID NO: 41 после очистки.
[00924] 3-2) После реакции 3-1) трижды промывали планшет 300 мкл PBS-T на лунку.
[00925] 4. Процесс реакции вторичных антител
[00926] 4-1) В качестве реакции вторичных антител добавляли 100 мкл пероксидазы хрена (HRP), конъюгированной с антителом против IgG мыши, распознающим антитело мыши, на лунку и давали им прореагировать при 37°C в течение одного часа.
[00927] 4-2) После реакции 4-1) трижды промывали планшет 300 мкл PBS-T на лунку.
[00928] Процесс подтверждения проявления цвета и поглощения
[00929] 5-1) Добавляли по 100 мкл раствора о-фенилендиамин дигидрохлорида (OPD) на лунку и давали прореагировать при 37°C в течение 10 минут, затем измеряли оптическую плотность при OD 450 нм (ридер микропланшетов Synergy HT, BioTek).
[00930] Получали титр антител в сыворотке путем преобразования коэффициента поглощения, который измеряли на основе концентрации 1 мг/мл моноклонального антитела против целевого антитела в качестве положительного контроля.
[00931] Экспериментальный пример 1.10. Подтверждение эффекта композиции для введения in vivo. 3. Подтверждение титра антител.
[00932] Экспериментальный метод подтверждения эффекта композиции для введения in vivo, введенной в экспериментальном примере 1.7, в отношении концентрации липидов в крови испытуемого заключается в следующем:
[00933] (1) Через неделю после введения каждой композиции собирали примерно 200 мкл крови из хвостовой вены испытуемого.
[00934] (2) Измерение концентрации триглицеридов (TG) в крови: использовали Triglyzyme-V (Shinyak Chemical Co., Ltd.). i) После смешивания 4 мкл образца крови и 300 мкл реагента для проявления цвета смесь подвергали реакции при 37°C в течение 5 минут.ii) Для полученного красного хинона измеряли оптическую плотность при 505 нм и рассчитывали концентрацию путем сравнения с концентрацией эталонного раствора.
[00935] (3) Измерение концентрации общего холестерина в крови: использовали Cholestezyme-V (Shinyak Chemical Co., Ltd.). i) После смешивания 4 мкл образца крови и 300 мкл реагента для проявления цвета смесь подвергали реакции при 37°C в течение 5 минут.ii) Для полученного красного хинона измеряли оптическую плотность при 505 нм и рассчитывали концентрацию путем сравнения с концентрацией эталонного раствора.
[00936] (4) Измерение концентрации липопротеинов высокой плотности (HDL) в крови: использовали HDL-C555 (Shinyak Chemical Co., Ltd.). i) После смешивания 10 мкл образца крови и 10 мкл реагента для преципитации смесь оставляли реагировать при комнатной температуре в течение не менее 10 минут. ii) Реагенты центрифугировали при 300 об/мин или выше и отделяли супернатант. iii) После смешивания 4 мкл супернатанта и 300 мкл реагента, проявляющего окраску, смесь реагировала при 37°C в течение 5 минут. iv) Для вышеуказанной реакции измеряли оптическую плотность при 555 нм и рассчитывали концентрацию путем сравнения с концентрацией эталонного раствора.
[00937] (5) Измерение концентрации липопротеинов низкой плотности (LDL) в крови. i) Реакцию проводили с использованием набора для прямого обнаружения холестерина LDL (Randox). ii) После реакции на стадии 2 для полученного хинона измеряли оптическую плотность при 600 нм и рассчитывали концентрацию путем сравнения с концентрацией эталонного раствора.
[00938] Экспериментальный пример 1.11. Подтверждение эффекта композиции для введения in vivo. 4. Подтверждение способности к липолизу и сравнение размера адипоцитов.
[00939] Способ подтверждения эффекта композиции для введения in vivo, введенной в экспериментальном примере 1.7, в отношении способности разрушать адипоциты и размер адипоцитов испытуемого с помощью гормончувствительной липазы (HSL) заключается в следующем:
[00940] 1. Разделение адипоцитов
[00941] 1-1) Разрезали ножницами эпидидимальную жировую ткань, добавляли 4 мл на 1 г KRB-буфера, включая 2% FBS, 2 мМ глюкозы и 1 мг/мл коллагеназы, встряхивали и давали им прореагировать при 37°C в течение одного часа при встряхивании.
[00942] 1-2) После завершения реакции пропускали полученную смесь через нейлоновую сетку 300 мкм, чтобы отфильтровать остатки жировой ткани и жировую ткань, а затем снова пропускали фильтрат через нейлоновую сетку 40 мкм, чтобы отделить адипоциты и макрофаги.
[00943] 1-3) Промывали адипоциты, отфильтрованные в 1-2), добавив DMEM с 10% FBS и 1% AA, и удаляли жидкость в нижнем слое с помощью шприца, чтобы получить адипоциты, из которых была удалена коллагеназа.
[00944] 2. Сравнение способности липолиза
[00945] 2-1) Высевали адипоциты, полученные в 1-3), в 48-луночный планшет в количестве 1×105 клеток/лунку и добавляли в общей сложности 1 мл DMEM (10% FBS, 1% АК) для культивирования при 37°С 5% CO2 в течение двух часов.
[00946] 2-2) Чтобы лунки индуцировали активность HSL, добавляли норадреналин до конечной концентрации 10-5 М.
[00947] 2-3) После завершения реакции 100 мкл супернатанта из каждой лунки давали прореагировать со 100 мкл свободного глицеринового реагента и измеряли оптическую плотность при 540 нм.
[00948] 3. Наблюдение за размером адипоцитов
[00949] 3-1) Высевали адипоциты, полученные в 1-3), в 48-луночный планшет в концентрации 1×105 клеток/мл на лунку, обрабатывали 10 мкМ DAPI, давали им прореагировать в течение двух часов и наблюдали под микроскопом.
[00950] 3-2) Чтобы подтвердить, являются ли клетки, окрашенные DAPI, адипоцитами, окрашивали липиды и ядра вместе и наблюдали. В частности, обрабатывали клетки 10 мкМ DAPI и 1:1000 HCS LipidTOX и давали им прореагировать в течение 24 часов, а затем наблюдали под микроскопом.
[00951] Экспериментальный пример 2, подтверждение эффекта пептида 1
[00952] Экспериментальный пример 2.1. Получение пептидов и эксперименты с ними.
[00953] После получения пептидов в соответствии с [Таблицей 1] в соответствии с экспериментальным примером 1.1 полученные пептиды подтверждали в соответствии с экспериментальными примерами 1.2-1.4. Композицию для введения in vivo, включающую пептиды согласно [Таблице 1], готовили в соответствии с экспериментальным примером 1.5.
[00954] [Таблица 1] Пептиды, использованные в экспериментальном примере 2, и композиция для введения in vivo, включающая их
[00955]
[00956] Испытуемый, показанный в [Таблице 2], был подготовлен в соответствии с экспериментальным примером 1.6.
[00957] [Таблица 2] Испытуемый, использованный в экспериментальном примере 2
[00958]
[00959] В соответствии с экспериментальным примером 1.7 испытуемому вводили композицию для введения in vivo. В частности, цикл введения был следующим: 7-недельный, 9-недельный, 12-недельный, 15-недельный и 18-недельный возраст.
[00960] Экспериментальный пример 2.2. Подтверждение экспериментальных результатов.
[00961] Чтобы подтвердить экспериментальные результаты экспериментального примера 2.1, массу испытуемого измеряли для каждой экспериментальной группы, указанной в [Таблице 2], в соответствии с экспериментальным примером 1.8.
[00962] Экспериментальные результаты показаны на ФИГ. 1-3.
[00963]
[00964] Экспериментальный пример 3, подтверждение эффекта пептида 2
[00965] Экспериментальный пример 3.1. Получение пептида и эксперимент с ним.
[00966] После приготовления пептидов в соответствии с [Таблицей 3] в соответствии с экспериментальным примером 1.1 приготовленные пептиды подтверждали в соответствии с экспериментальными примерами 1.2-1.4. Готовили композицию для введения in vivo, включающую пептиды согласно [Таблице 3] в соответствии с экспериментальным примером 1.5.
[00967] [Таблица 3] Пептиды, использованные в экспериментальном примере 3, и композиция для введения in vivo, включающая их
[00968]
[00969] Испытуемый, показанный в [Таблице 4], был подготовлен в соответствии с экспериментальным примером 1.6.
[00970] [Таблица 4] Испытуемый, использованный в экспериментальном примере 3
[00971]
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
[00972] В частности, Lean обозначает контрольную группу с нормальной массой, Obese обозначает группу с ожирением, вызванным диетой с высоким содержанием жиров, а Mock обозначает группу, получающую плацебо (то же самое в дальнейшем).
[00973] В соответствии с экспериментальным примером 1.7 испытуемому вводили композицию для введения in vivo. В частности, цикл введения был следующим: 8-недельный, 10-недельный, 12-недельный и 14-недельный возраст.
[00974] Экспериментальный пример 3.2. Подтверждение экспериментальных результатов.
[00975] Чтобы подтвердить экспериментальные результаты экспериментального примера 3.1, следующие эксперименты проводили на испытуемом для каждой экспериментальной группы, описанной в [Таблице 4].
[00976] (1) В соответствии с экспериментальным примером 1.8 измеряли массу испытуемого для каждой экспериментальной группы.
[00977] (2) В соответствии с экспериментальным примером 1.9 подтверждали титры антител, наблюдаемые у испытуемого для каждой экспериментальной группы.
[00978] (3) В соответствии с экспериментальным примером 1.10 измеряли концентрацию липидов в крови испытуемого для каждой экспериментальной группы.
[00979] (4) В соответствии с экспериментальным примером 1.11 подтверждали способность испытуемого к липолизу для каждой экспериментальной группы и наблюдался размер адипоцитов.
[00980] Экспериментальные результаты показаны на ФИГ. 4-9.
[00981]
[00982] Экспериментальный пример 4, подтверждение эффекта пептида 3
[00983] Экспериментальный пример 4.1. Получение пептида и эксперимент с ним.
[00984] После приготовления пептидов в соответствии с [Таблицей 5] в соответствии с экспериментальным примером 1.1 приготовленные пептиды подтверждали в соответствии с экспериментальными примерами 1.2-1.4. Готовили композицию для введения in vivo, включающую пептиды согласно [Таблице 5] в соответствии с экспериментальным примером 1.5.
[00985] [Таблица 5] Пептиды, использованные в экспериментальном примере 4, и композиция для введения in vivo, включающая их
[00986]
[00987] В частности, в примере 4 конечная концентрация пептида составляла 30 мкг/100 мкл.
[00988] Испытуемый, показанный в [Таблице 6], был подготовлен в соответствии с экспериментальным примером 1.6.
[00989] [Таблица 6] Испытуемый, использованный в экспериментальном примере 4
[00990]
через 11 недель: диета с высоким содержанием жира
через 11 недель: диета с высоким содержанием жира
через 11 недель: диета с высоким содержанием жира
через 11 недель: диета с высоким содержанием жира
через 11 недель: диета с высоким содержанием жира
[00991] В соответствии с экспериментальным примером 1.7 испытуемому вводили композицию для введения in vivo. В частности, цикл введения был следующим: 8-недельный, 11-недельный, 14-недельный, 17-недельный и 20-недельный возраст.
[00992] Экспериментальный пример 4.2. Подтверждение экспериментальных результатов.
[00993] Чтобы подтвердить экспериментальные результаты экспериментального примера 4.1, следующие эксперименты проводили на испытуемом для каждой экспериментальной группы, описанной в [Таблица 6].
[00994] (1) В соответствии с экспериментальным примером 1.8 измеряли массу испытуемого для каждой экспериментальной группы.
[00995] (2) В соответствии с экспериментальным примером 1.9 подтверждали титры антител, наблюдаемые у испытуемого для каждой экспериментальной группы.
[00996] Экспериментальные результаты показаны на ФИГ. 10 и 11.
[00997]
[00998] Экспериментальный пример 5, подтверждение эффекта пептида 4
[00999] Экспериментальный пример 5.1. Получение пептида и эксперимент с ним.
[001000] После приготовления пептидов в соответствии с[Таблицей 7] в соответствии с экспериментальным примером 1.1 приготовленные пептиды подтверждали в соответствии с экспериментальными примерами 1.2-1.4. Готовили композицию для введения in vivo, включающую пептиды согласно [Таблице 7] в соответствии с экспериментальным примером 1.5.
[001001] [Таблица 7] Пептиды, использованные в экспериментальном примере 5, и композиция для введения in vivo, включающая их
[001002]
[001003] Испытуемый, показанный в [Таблице 8], был подготовлен в соответствии с экспериментальным примером 1.6.
[001004] [Таблица 8] Испытуемый, использованный в экспериментальном примере 5
[001005]
[001006] В соответствии с экспериментальным примером 1.7 испытуемому вводили композицию для введения in vivo. В частности, цикл введения был следующим: 11-недельный, 13-недельный, 15-недельный и 17-недельный возраст.
[001007] Экспериментальный пример 5.2. Подтверждение экспериментальных результатов.
[001008] Чтобы подтвердить экспериментальные результаты экспериментального примера 5.1, массу испытуемого измеряли для каждой экспериментальной группы, указанной в[Таблице 8], в соответствии с экспериментальным примером 1.8.
[001009] Экспериментальные результаты показаны на фиг.12.
[001010]
[001011] Экспериментальный пример 6, подтверждение эффекта пептида 5
[001012] Экспериментальный пример 6.1. Получение пептида и эксперимент с ним.
[001013] После приготовления пептидов в соответствии с [Таблицей 9] в соответствии с экспериментальным примером 1.1 приготовленные пептиды подтверждали в соответствии с экспериментальными примерами 1.2-1.4. Готовили композицию для введения in vivo, включающую пептиды согласно [Таблице 9] в соответствии с экспериментальным примером 1.5.
[001014] [Таблица 9] Пептиды, использованные в экспериментальном примере 6, и композиция для введения in vivo, включающая их
[001015]
[001016] Испытуемый, показанный в [Таблице 10], был подготовлен в соответствии с экспериментальным примером 1.6.
[001017] [Таблица 10] Испытуемый, использованный в экспериментальном примере 6
[001018]
+))
через 10 недель: диета с высоким содержанием жира
(+/-))
через 10 недель: диета с высоким содержанием жира
(-/-))
Homo(-/-)
через 10 недель: диета с высоким содержанием жира
[001019] *Мышь C57BL/6J-Rag2em1hwl/Korl представляет собой мышь, у которой нокаутирован ген Rag2, который является геном, участвующим в способности мыши C57BL/6J продуцировать антитела.
[001020] **«Wild» (+/+) относится к мыши дикого типа, у которой ни один ген не мутирован, «Hetero» (+/-) относится к гетерозиготной мыши, а «Homo» (-/-) относится к гомозиготной мыши.
[001021] ***Homo (-/-) мыши не обладают способностью вырабатывать антитела в организме.
[001022] В соответствии с экспериментальным примером 1.7 испытуемому вводили композицию для введения in vivo. В частности, цикл введения был следующим: 8-недельный, 10-недельный, 12-недельный и 14-недельный возраст.
[001023] Экспериментальный пример 6.2. Подтверждение экспериментальных результатов.
[001024] Чтобы подтвердить экспериментальные результаты Экспериментального примера 6.1, массу испытуемого измеряли для каждой экспериментальной группы, указанной в [Таблице 10], в соответствии с Экспериментальным примером 1.8.
[001025] Экспериментальные результаты показаны на ФИГ. 13.
[001026]
[001027] Экспериментальный пример 7. Подтверждение эффекта пептида 6.
[001028] Экспериментальный пример 7.1. Получение пептида и эксперимент с ним.
[001029] После приготовления пептидов в соответствии с [Таблицей 11] в соответствии с экспериментальным примером 1.1 приготовленные пептиды подтверждали в соответствии с экспериментальными примерами 1.2-1.4. Готовили композицию для введения in vivo, включающую пептиды согласно [Таблице 11] в соответствии с экспериментальным примером 1.5.
[001030] [Таблица 11] Пептиды, использованные в экспериментальном примере 7, и композиция для введения in vivo, включающая их
[001031]
[001032] Испытуемый, показанный в [Таблице 12], был подготовлен в соответствии с экспериментальным примером 1.6.
[001033] [Таблица 12] Испытуемый, использованный в экспериментальном примере 7
[001034]
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
[001035] В соответствии с экспериментальным примером 1.7 испытуемому вводили композицию для введения in vivo. В частности, цикл введения был следующим: 8-недельный, 10-недельный, 12-недельный и 14-недельный возраст.
[001036] Экспериментальный пример 7.2. Подтверждение экспериментальных результатов.
[001037] Чтобы подтвердить экспериментальные результаты экспериментального примера 7.1, следующие эксперименты были проведены на испытуемом для каждой экспериментальной группы, описанной в [Таблице 12].
[001038] (1) В соответствии с экспериментальным примером 1.8 измеряли массу испытуемого для каждой экспериментальной группы.
[001039] (2) В соответствии с экспериментальным примером 1.9 были подтверждены титры антител, наблюдаемые у испытуемого для каждой экспериментальной группы через 11 недель, 16 недель и 19 недель. В частности, целевой антиген определяли с использованием RNVPPIFNDVYWIAF (SEQ ID NO: 6).
[001040] Экспериментальные результаты показаны на ФИГ. 14-25.
[001041] В результате эксперимента все экспериментальные группы от 6-1 (P1) до экспериментальных групп 6-9 (P9) продемонстрировали отчетливый эффект потери массы по сравнению с контрольной группой (с ожирением), которые получали диету с высоким содержанием жира.
[001042] Результаты теста на титр антител по экспериментальной группе можно интерпретировать следующим образом:
[001043] Поскольку контрольным группам (то есть Lean и Obese) антиген не вводили, титры антител не отображались.
[001044] Принимая во внимание план эксперимента, в экспериментальных группах в основном подтверждали титры антител к SEQ ID NO: 6, включенных в используемые пептиды. Хотя титры антител к последовательностям, отличным от SEQ ID NO: 6, включенным в используемые пептиды, не были подтверждены по отдельности, эффект потери массы, обусловленный гуморальным иммунитетом, подтвержден вместе с ними.
[001045] Конкретные результаты будут описаны ниже.
[001046] В случае экспериментальной группы 6-1 (P1) были индивидуумы, демонстрирующие высокий титр антител в зависимости от индивидуума, а также были индивидуумы, у которых титр антител отсутствовал. Эти результаты можно интерпретировать следующим образом: у некоторых индивидуумов экспериментальной группы 6-1 (P1) был индуцирован гуморальный иммунитет к RNVPPIFNDVYWIAF (SEQ ID NO: 6), включенному в пример 6, таким образом демонстрируя как эффект потери массы, так и наблюдаемый титр антитела; и хотя у некоторых индивидуумов индуцировался гуморальный иммунитет к CRFRGLISLSQVYLS (SEQ ID NO: 7), включенному в пример 6, что приводило к потере массы, антиген, используемый в твердофазном иммуноферментном анализе (ИФА), состоял только из RNVPPIFNDVYWIAF (SEQ ID NO:6) и отсутствовал другой антиген (т.е. антиген SEQ ID NO:7), следовательно, титр антител по вышеуказанному эксперименту не наблюдался даже в присутствии антитела.
[001047] В случае экспериментальной группы 6-2 (P2) наблюдали титр антител к RNVPPIFNDVYWIAF (SEQ ID NO: 6), включенному в пример 7, и появился эффект потери массы, это можно интерпретировать так, что В-клеточный эпитоп, включенный в пептид примера 7, хорошо индуцировал к нему гуморальный иммунитет.
[001048] В случае экспериментальной группы 6-3 (P3) наблюдали титр антител к RNVPPIFNDVYWIAF (SEQ ID NO: 6), включенному в пример 8, и появился эффект потери массы, что можно интерпретировать так, что В-клеточный эпитоп включенный в состав пептида примера 8, хорошо индуцировал к нему гуморальный иммунитет.
[001049] В случае экспериментальной группы 6-4 (P4) можно интерпретировать, что был индуцирован гуморальный иммунитет к KTTKQSFDLSVKAQYKKNKH (SEQ ID NO: 8) и/или CRFRGLISLSQVYLS (SEQ ID NO: 7), включенным в пример 9, таким образом, титры антитела против RNVPPIFNDVYWIAF (SEQ ID NO: 6) не были показаны, в то время как наблюдался эффект потери массы.
[001050] Эти результаты можно интерпретировать так, что у некоторых индивидуумов экспериментальной группы 6-5 (P5) был индуцирован гуморальный иммунитет против RNVPPIFNDVYWIAF (SEQ ID NO: 6), включенного в пример 10, так что наблюдали как эффект потери массы, так и титр антител, у других особей экспериментальной группы 6-5 (P5) индуцировался гуморальный иммунитет против CRFRGLISLSQVYLS (SEQ ID NO: 7), включенного в пример 10, так что наблюдали эффект потери массы, но титр антител согласно приведенному выше эксперименту не наблюдали.
[001051] В случае экспериментальной группы 6-7 (P7), несмотря на некоторые различия между индивидуумами, наблюдали антитела к RNVPPIFNDVYWIAF (SEQ ID NO: 6) и проявлялся эффект потери массы, следовательно, это можно интерпретировать так, что B-клеточный эпитоп, включенный в состав пептида Примера 11, хорошо индуцировал гуморальный иммунитет.
[001052] В случае экспериментальной группы 6-8 (P8), поскольку был показан титр антител против RNVPPIFNDVYWIAF (SEQ ID NO: 6), включенного в пример 12, и был показан эффект потери массы, это можно интерпретировать так, что В-клеточный эпитоп, включенный в пептид Примера 12, хорошо индуцировал гуморальный иммунитет.
[001053] В случае экспериментальной группы 6-9 (P9) были некоторые различия между индивидуумами. Однако, поскольку наблюдали антитело против RNVPPIFNDVYWIAF (SEQ ID NO: 6) и проявлялся эффект потери массы, то это можно интерпретировать так, что В-клеточный эпитоп, включенный в пептид примера 13, хорошо индуцировал гуморальный иммунитет.
[001054]
[001055] Экспериментальный пример 8, подтверждение эффекта пептида 7
[001056] Экспериментальный пример 8.1. Получение пептида и эксперимент с ним.
[001057] После приготовления пептидов в соответствии с [Таблицей 13] в соответствии с экспериментальным примером 1.1 полученные пептиды подтверждали в соответствии с экспериментальными примерами 1.2-1.4. Готовили композицию для введения in vivo, включающую пептиды согласно [Таблице 13] в соответствии с экспериментальным примером 1.5.
[001058] [Таблица 13] Пептиды, использованные в экспериментальном примере 8, и композиция для введения in vivo, включающая их
[001059]
[001060] Испытуемый, показанный в [Таблице 14], подготовлен в соответствии с экспериментальным примером 1.6.
[001061] [Таблица 14] Испытуемый, использованный в экспериментальном примере 8
[001062]
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
[001063] В соответствии с экспериментальным примером 1.7 композицию для введения in vivo вводили испытуемому. В частности, цикл введения, время введения и частота введения могут быть надлежащим образом изменены в соответствии с планом эксперимента. Например, композицию можно вводить четыре раза с интервалом в две недели испытуемому в возрасте 8 недель, но не ограничиваясь этим.
[001064] Вышеупомянутый экспериментальный метод может быть надлежащим образом модифицирован по мере необходимости.
[001065] Экспериментальный пример 8.2. Подтверждение экспериментальных результатов.
[001066] Чтобы подтвердить экспериментальные результаты Экспериментального примера 8.1, следующие эксперименты проводили на испытуемом для каждой экспериментальной группы, описанной в [Таблице 14].
[001067] (1) Согласно экспериментальному примеру 1.8 измеряли массу испытуемого для каждой экспериментальной группы.
[001068] (2) В соответствии с экспериментальным примером 1.9 подтверждали титры антител, наблюдаемые у испытуемого для каждой экспериментальной группы.
[001069] (3) В соответствии с экспериментальным примером 1.10 измеряли концентрацию липидов в крови испытуемого для каждой экспериментальной группы.
[001070] (4) В соответствии с экспериментальным примером 1.11 подтверждали способность испытуемого к липолизу для каждой экспериментальной группы и наблюдали размер адипоцитов.
[001071]
[001072] Экспериментальный пример 9, подтверждение эффекта пептида 8
[001073] Экспериментальный пример 9.1. Получение пептида и эксперимент с ним.
[001074] В соответствии с экспериментальным примером 1.1 получали пептид, представленный одной или более последовательностями, выбранными из группы, состоящей из следующих: SEQ ID NO: 56-158, SEQ ID NO: 160-161 и SEQ ID NO: 198-220.
[001075] Полученные пептиды подтверждали согласно экспериментальным примерам 1.2-1.4. В соответствии с экспериментальным примером 1.5 готовили композицию для введения in vivo, включающую полученные пептиды.
[001076] В частности, пептид может быть получен путем выбора только некоторых последовательностей из приведенных выше групп последовательностей, и при необходимости могут быть проведены повторные эксперименты с несколькими комбинациями.
[001077] Испытуемого для приготовленных пептидов готовили в соответствии с экспериментальным примером 1.6. В частности, примеры используемых контрольных и экспериментальных групп показаны в [Таблице 15], и каждая экспериментальная группа определяется в соответствии с условиями Экспериментального примера 1.6 и [Таблица 15].
[001078] [Таблица 15] Испытуемый, использованный в экспериментальном примере 9
[001079]
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
через 10 недель: диета с высоким содержанием жира
[001080] В частности, готовили столько экспериментальных групп, сколько приготовлено пептидов.
[001081] Согласно экспериментальному примеру 1.7 композицию для введения in vivo вводили испытуемому. В частности, цикл введения, время введения и частота введения могут быть надлежащим образом изменены в соответствии с планом эксперимента. Например, композицию можно вводить четыре раза с интервалом в две недели испытуемому в возрасте 8 недель, но не ограничиваясь этим.
[001082] Вышеупомянутый экспериментальный метод может быть надлежащим образом модифицирован по мере необходимости.
[001083] Экспериментальный пример 9.2. Подтверждение экспериментальных результатов.
[001084] Чтобы подтвердить экспериментальные результаты экспериментального примера 9.1, следующие эксперименты проводили на испытуемом для каждой экспериментальной группы.
[001085] (1) Согласно экспериментальному примеру 1.8 измеряли массу испытуемого для каждой экспериментальной группы.
[001086] (2) В соответствии с экспериментальным примером 1.9 подтверждали титры антител, наблюдаемые у испытуемого для каждой экспериментальной группы.
[001087] (3) В соответствии с экспериментальным примером 1.10 измеряли концентрацию липидов в крови испытуемого для каждой экспериментальной группы.
[001088] (4) В соответствии с экспериментальным примером 1.11 подтверждали способность к липолизу испытуемого в каждой экспериментальной группе и наблюдали размер адипоцитов.
[001089]
[001090] Экспериментальный пример 10, подтверждение эффекта пептида 9
[001091] Экспериментальный пример 10.1. Получение пептида и эксперимент с ним.
[001092] После приготовления пептидов в соответствии с примерами 81-90 в разделе «Возможные примеры изобретения» в соответствии с экспериментальным примером 1.1 полученные пептиды подтверждали в соответствии с экспериментальными примерами 1.2-1.4. В соответствии с экспериментальным примером 1.5 готовили композицию для введения in vivo, включающую полученные пептиды.
[001093] В частности, могут быть отобраны и приготовлены только части пептидов в соответствии с примерами 81-90 раздела «Возможные примеры изобретения», и при необходимости можно повторить эксперименты с множеством комбинаций.
[001094] Испытуемого для приготовленного пептида готовили в соответствии с экспериментальным примером 1.6. В частности, используемые контрольные и экспериментальные группы определяли в соответствии с условиями экспериментального примера 1.6 и [Таблицы 15].
[001095] В соответствии с экспериментальным примером 1.7 композицию для введения in vivo вводили испытуемому. В частности, цикл введения, время введения и частота введения могут быть надлежащим образом изменены в соответствии с планом эксперимента. Например, композицию можно вводить четыре раза с интервалом в две недели испытуемому в возрасте 8 недель, но не ограничиваясь этим.
[001096] Вышеупомянутый экспериментальный метод может быть надлежащим образом модифицирован по мере необходимости.
[001097] Экспериментальный пример 10.2. Подтверждение экспериментальных результатов.
[001098] Чтобы подтвердить экспериментальные результаты экспериментального примера 10.1, следующие эксперименты проводили на испытуемом для каждой экспериментальной группы.
[001099] (1) Согласно экспериментальному примеру 1.8 измеряли массу испытуемого для каждой экспериментальной группы.
[001100] (2) В соответствии с экспериментальным примером 1.9 подтверждали титры антител, наблюдаемые у испытуемого для каждой экспериментальной группы.
[001101] (3) В соответствии с экспериментальным примером 1.10 измеряли концентрацию липидов в крови испытуемого для каждой экспериментальной группы.
[001102] (4) В соответствии с экспериментальным примером 1.11 подтверждали способность испытуемого к липолизу для каждой экспериментальной группы и наблюдали размер адипоцитов.
[001103]
Промышленная применимость
[001104] Пептидная единица, пептид и/или нуклеиновая кислота, кодирующая их, предложенные в настоящем описании, могут быть использованы для получения иммунотерапевтического средства, в частности, средства для лечения ожирения, и иммунотерапевтическое средство может проявлять терапевтический эффект путем создания предполагаемого гуморального иммунитета при введении в тело пациента.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3"
fileName="P220107US_Sequence_Listing_reviesed_01.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2022-11-08">
<ApplicationIdentification>
<IPOfficeCode>US</IPOfficeCode>
<ApplicationNumberText>17/823,180</ApplicationNumberText>
<FilingDate>2022-08-30</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>P220107US</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>10-2020-0091033</ApplicationNumberText>
<FilingDate>2020-07-22</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ko">(주)쓰리에이치바이오</ApplicantName>
<ApplicantNameLatin>3HBio</ApplicantNameLatin>
<InventionTitle languageCode="en">A PEPTIDE USED FOR
IMMUNOTHERAPEUTICS</InventionTitle>
<SequenceTotalQuantity>309</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q881">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2nd amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q883">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q882">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q885">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q884">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PKYVKQNTLKLAT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q887">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q886">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILMQYIKANSKFIGI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q889">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q888">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSIALSSLMVAQAIP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q891">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q890">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q893">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q892">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q895">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q894">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q897">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q896">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q899">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q898">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q901">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q900">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQ</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>12</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..12</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q903">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q902">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>12</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..12</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q905">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q904">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYW</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>12</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..12</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q907">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q906">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q909">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q908">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q911">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q910">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q913">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q912">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q915">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q914">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q917">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q916">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PIFNDVYWIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q919">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q918">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q921">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q920">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSFDLSVKAQYKKNKH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>12</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..12</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q923">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q922">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PPIFNDVYWIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>12</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..12</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q925">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q924">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RGLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q927">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q926">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KQSFDLSVKAQYKKNKH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q929">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q928">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VPPIFNDVYWIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q931">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q930">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FRGLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q933">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q932">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TKQSFDLSVKAQYKKNKH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q935">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q934">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>NVPPIFNDVYWIA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q937">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q936">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RFRGLISLSQVYL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q939">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q938">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TKQSFDLSVKAQYKKN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q941">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q940">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VPPIFNDVYWI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q943">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q942">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FRGLISLSQVY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q945">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q944">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TKQSFDLSVKAQYKKN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q947">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q946">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PPIFNDVYW</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q949">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q948">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RGLISLSQV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>14</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q951">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q950">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KQSFDLSVKAQYKK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>4</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2048">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q953">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>4</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q955">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q954">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>WIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>4</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2042">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q957">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFR</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>4</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q959">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q958">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length/>
<INSDSeq_moltype/>
<INSDSeq_division/>
<INSDSeq_sequence>000</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2049">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Auxiliary part</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q961">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GSHHHHHHGSDDDDK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>6</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q963">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Auxiliary part</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q962">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>HHHHHH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q965">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>His-tag sequence</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q964">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVD</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q967">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Linker peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q966">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GGGGSGGGGGGSS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q969">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q968">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q970">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q971">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q972">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q973">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q974">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q975">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>1st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q977">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q976">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q978">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2nd aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q979">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>4th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q980">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>14th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q981">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>15th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXKXVAAWTLKAAXXRNVPPIFNDVYWIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="58">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q982">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q984">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q983">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q985">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q986">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q987">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q988">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLSXXKXVAAWTLKAAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="59">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q989">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>1st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q991">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q990">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q992">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2nd aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q993">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>4th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q994">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>14th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q995">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>15th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXKXVAAWTLKAAXXCRFRGLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="60">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q996">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>21st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q998">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q997">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q999">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>22nd aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1000">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1001">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1002">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>35th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXX</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="61">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1003">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>1st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1005">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1004">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1006">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2nd aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1007">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>4th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1008">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>14th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1009">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>15th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXKXVAAWTLKAAXXKTTKQSFDLSVKAQYKKNKH</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="62">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1010">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1012">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1011">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFKXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="63">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1013">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2nd amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1015">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1014">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAAWTLKAARNVPPIFNDVYWIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="64">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1016">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>13rd amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1018">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1017">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYKXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="65">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1019">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>13rd amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1021">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1020">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PIFNDVYWIAFKXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="66">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1022">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>11st amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1024">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1023">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PPIFNDVYWKXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="67">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1025">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1027">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1026">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFKXVAAWTLKAAHHHHHH</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="68">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1028">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1030">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1029">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1031">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1032">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1033">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1034">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXGSHHHHHHGSDDDDK</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="69">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1035">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1037">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1036">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1038">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1039">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1040">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1041">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GSHHHHHHGSDDDDKXXKXVAAWTLKAAXXRNVPPIFNDVYWIAF</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="70">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1042">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>31st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1044">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1043">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1045">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>32nd aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1046">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1047">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>44th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1048">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>45th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFGSHHHHHHGSDDDDKXXKXVAAWTLKAAXX</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="71">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1049">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1051">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1050">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1052">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1053">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1054">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1055">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GSHHHHHHGSDDDDKXXKXVAAWTLKAAXXCRFRGLISLSQVYLS</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="72">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1057">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1056">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1058">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>31st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1059">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>32nd aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1060">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1061">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>44th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1062">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>45th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GSHHHHHHGSDDDDKCRFRGLISLSQVYLSXXKXVAAWTLKAAXX</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="73">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1064">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1063">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1065">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1066">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1067">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1068">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1069">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLSXXKXVAAWTLKAAXXGSHHHHHHGSDDDDK</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="74">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1071">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1070">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1072">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1073">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1074">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1075">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1076">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GSHHHHHHGSDDDDKXXKXVAAWTLKAAXXKTTKQSFDLSVKAQYKKNK
H</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="75">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1078">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1077">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1079">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>36th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1080">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>37th aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1081">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>39th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1082">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1083">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GSHHHHHHGSDDDDKKTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAX
X</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="76">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1085">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1084">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1086">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>21st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1087">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>22nd aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1088">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1089">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1090">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>35th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXGSHHHHHHGSDDDD
K</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="77">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1092">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1091">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1093">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>36th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1094">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>37th aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1095">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>39th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1096">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1097">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFXXKXVAAWTLKAAX
X</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="78">
<INSDSeq>
<INSDSeq_length>65</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1099">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1098">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>51</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1100">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>51st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>52</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1101">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>52nd aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>54</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1102">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>54th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>64</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1103">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>64th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1104">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>65th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDGSHHHHHHGSDDDDKRNVPPIFNDVYWIA
FXXKXVAAWTLKAAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="79">
<INSDSeq>
<INSDSeq_length>65</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1106">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1105">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1107">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>36th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1108">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>37th aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1109">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>39th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1110">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1111">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFXXKXVAAWTLKAAX
XGSHHHHHHGSDDDDK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="80">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1113">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1112">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFILMQYIKANSKFIGI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="81">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1115">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1114">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFILMQYIKANSKFIGIPMGLPQSIALSSLMVAQ</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="82">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1117">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1116">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1118">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1119">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>18th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1120">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>20th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1121">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1122">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>31st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRNVPPIFNDVYWIAFXXKXVAAWTLKAAXXC</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="83">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1124">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1123">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1125">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>31st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1126">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>32nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1127">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1128">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>44th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1129">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>45th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFRNVPPIFNDVYWIAFXXKXVAAWTLKAAXX</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="84">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1131">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1130">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1132">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>31st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1133">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>32nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1134">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1135">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>44th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1136">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>45th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFCRFRGLISLSQVYLSXXKXVAAWTLKAAXX</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="85">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1138">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1137">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1139">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>36th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1140">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>37th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1141">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>39th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1142">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1143">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFKTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAX
X</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="86">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1146">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1145">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q2043">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>31st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q2044">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>32nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q2045">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q2046">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>44th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q2047">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>45th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLSRNVPPIFNDVYWIAFXXKXVAAWTLKAAXX</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="87">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1148">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1147">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1149">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>31st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1150">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>32nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1151">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1152">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>44th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1153">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>45th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLSCRFRGLISLSQVYLSXXKXVAAWTLKAAXX</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="88">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1155">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1154">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1156">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>36th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1157">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>37th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1158">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>39th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1159">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1160">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLSKTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAX
X</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="89">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1162">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1161">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1163">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>36th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1164">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>37th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1165">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>39th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1166">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1167">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHRNVPPIFNDVYWIAFXXKXVAAWTLKAAX
X</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="90">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1169">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1168">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1170">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>36th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1171">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>37th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1172">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>39th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1173">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1174">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHCRFRGLISLSQVYLSXXKXVAAWTLKAAX
X</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="91">
<INSDSeq>
<INSDSeq_length>55</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..55</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1176">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1175">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1177">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>41st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1178">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>42nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1179">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>44th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>54</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1180">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>54th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>55</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1181">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>55th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHKTTKQSFDLSVKAQYKKNKHXXKXVAAWT
LKAAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="92">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1183">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1182">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1184">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>32nd amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFCRFRGLISLSQVYLSKXVAAWTLKAA</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="93">
<INSDSeq>
<INSDSeq_length>33</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1186">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1185">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1187">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PIFNDVYWIAFGLISLSQVYLSKXVAAWTLKAA</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="94">
<INSDSeq>
<INSDSeq_length>33</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1189">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1188">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1190">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYCRFRGLISLSQKXVAAWTLKAA</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="95">
<INSDSeq>
<INSDSeq_length>33</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1192">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1191">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1193">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PIFNDVYWIAFCRFRGLISLSQKXVAAWTLKAA</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="96">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1195">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1194">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1196">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>20th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PPIFNDVYWRGLISLSQVKXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="97">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1198">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1197">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1199">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>32nd amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFCRFRGLISLSQVYLSKXVAAWTLKAAHHHHHH</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="98">
<INSDSeq>
<INSDSeq_length>65</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1201">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1200">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>51</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1202">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>51st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>52</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1203">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>52nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>54</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1204">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>54th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>64</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1205">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>64th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1206">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>65th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFCRFRGLISLSQVYL
SXXKXVAAWTLKAAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="99">
<INSDSeq>
<INSDSeq_length>63</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1208">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1207">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>51</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1209">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>51st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>52</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1210">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>52nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>54</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1211">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>54th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFCRFRGLISLSQVYL
SXXKXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="100">
<INSDSeq>
<INSDSeq_length>43</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1213">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1212">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1214">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>31st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1215">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>32nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1216">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFCRFRGLISLSQVYLSXXKXVAAWTLKAA</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="101">
<INSDSeq>
<INSDSeq_length>76</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..76</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1218">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1217">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>64</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1219">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>64th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1220">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>65th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>67</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1221">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>67th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSR
NVPPIFNDVYWIAFXXKXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="102">
<INSDSeq>
<INSDSeq_length>56</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..56</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1223">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1222">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1224">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>44th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1225">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>45th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1226">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>47th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFGGGGSGGGGGGSSRNVPPIFNDVYWIAFXXKXVA
AWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="103">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1228">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1227">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1229">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>44th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1230">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>45th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1231">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>47th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>57</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1232">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>57th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1233">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>58th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFGGGGSGGGGGGSSRNVPPIFNDVYWIAFXXKXVA
AWTLKAAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="104">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1235">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1234">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFRNVPPIFNDVYWIAFILMQYIKANSKFIGI</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="105">
<INSDSeq>
<INSDSeq_length>62</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..62</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1237">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1236">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFRNVPPIFNDVYWIAFILMQYIKANSKFIGIPMGL
PQSIALSSLMVAQ</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="106">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1239">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1238">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1240">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>32nd amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1241">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>33rd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1242">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>35th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1243">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>45th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1244">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>46th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRNVPPIFNDVYWIAFCRFRGLISLSQVYLSXXKXVAAWTLKAAXXC</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="107">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1246">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1245">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1247">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1248">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1249">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1250">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1251">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXRNVPPIFNDVYWIAF</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="108">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1253">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1252">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1254">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1255">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1256">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1257">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1258">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXCRFRGLISLSQVYLS</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="109">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1260">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1259">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1261">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1262">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1263">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1264">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1265">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXKTTKQSFDLSVKAQYKKNK
H</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="110">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1267">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1266">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1268">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1269">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1270">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1271">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1272">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLSXXKXVAAWTLKAAXXRNVPPIFNDVYWIAF</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="111">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1274">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1273">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1275">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1276">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1277">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1278">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1279">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLSXXKXVAAWTLKAAXXCRFRGLISLSQVYLS</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="112">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1281">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1280">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1282">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1283">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1284">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1285">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1286">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXKTTKQSFDLSVKAQYKKNK
H</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="113">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1288">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1287">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1289">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>21st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1290">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>22nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1291">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1292">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1293">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>35th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXRNVPPIFNDVYWIA
F</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="114">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1295">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1294">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1296">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>21st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1297">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>22nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1298">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1299">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1300">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>35th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXCRFRGLISLSQVYL
S</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="115">
<INSDSeq>
<INSDSeq_length>55</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..55</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1302">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1301">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1303">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>21st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1304">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>22nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1305">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1306">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1307">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>35th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXKTTKQSFDLSVKAQ
YKKNKH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="116">
<INSDSeq>
<INSDSeq_length>33</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1309">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1308">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1310">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>13rd amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PIFNDVYWIAFKXVAAWTLKAACRFRGLISLSQ</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="117">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1312">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1311">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1313">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa can be any naturally occurring amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PPIFNDVYWKXVAAWTLKAARGLISLSQV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="118">
<INSDSeq>
<INSDSeq_length>65</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1315">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1314">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1316">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>36th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1317">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>37th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1318">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>39th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1319">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1320">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFXXKXVAAWTLKAAX
XRNVPPIFNDVYWIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="119">
<INSDSeq>
<INSDSeq_length>108</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1322">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1321">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSI
LMQYIKANSKFIGIPMGLPQSIALSSLMVAQGGGGSGGGGGGSSCRFRGLISLSQVYLS</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="120">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1324">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1323">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFILMQYIKANSKFIGICRFRGLISLSQVYLS</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="121">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1326">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1325">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1327">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1328">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1329">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1330">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1331">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXPKYVKQNTLKLATXCRFRGLISLSQVYLS</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="122">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1333">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1332">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1334">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1335">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>18th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1336">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>20th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1337">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1338">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>31st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRNVPPIFNDVYWIAFXXKXVAAWTLKAAXXRNVPPIFNDVYWIAFC</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="123">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1340">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1339">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1341">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1342">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1343">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1344">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1345">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGI</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="124">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1347">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1346">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1348">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1349">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1350">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1351">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1352">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLSXXKXVAAWTLKAAXXILMQYIKANSKFIGI</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="125">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1354">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1353">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1355">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>21st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1356">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>22nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1357">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1358">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1359">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>35th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXILMQYIKANSKFIG
I</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="126">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1361">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1360">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1362">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1363">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1364">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1365">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1366">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILMQYIKANSKFIGIXXKXVAAWTLKAAXXRNVPPIFNDVYWIAF</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="127">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1368">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1367">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1369">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1370">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1371">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1372">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1373">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILMQYIKANSKFIGIXXKXVAAWTLKAAXXCRFRGLISLSQVYLS</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="128">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1375">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1374">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1376">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1377">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1378">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1379">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1380">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILMQYIKANSKFIGIXXKXVAAWTLKAAXXKTTKQSFDLSVKAQYKKNK
H</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="129">
<INSDSeq>
<INSDSeq_length>33</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1382">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1381">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1383">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>13rd amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1384">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa)is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PIFNDVYWIAFKXVAAWTLKAAKXVAAWTLKAA</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="130">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1386">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1385">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1387">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>11st amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1388">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>21st amino acid(Xaa)is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PPIFNDVYWKXVAAWTLKAAKXVAAWTLKAA</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="131">
<INSDSeq>
<INSDSeq_length>65</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1390">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1389">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1391">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>36th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1392">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>37th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1393">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>39th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1394">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1395">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFXXKXVAAWTLKAAX
XILMQYIKANSKFIGI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="132">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1397">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1396">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1398">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>36th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1399">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>37th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1400">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>39th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1401">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1402">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDILMQYIKANSKFIGIXXKXVAAWTLKAAX
X</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="133">
<INSDSeq>
<INSDSeq_length>78</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..78</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1404">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1403">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1405">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1406">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>52</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1407">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>52nd amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>62</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1408">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>62nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1409">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>63rd amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSX
XKXVAAWTLKAAXXILMQYIKANSKFIGI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="134">
<INSDSeq>
<INSDSeq_length>125</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..125</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1411">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1410">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSI
LMQYIKANSKFIGIPMGLPQSIALSSLMVAQGGGGSGGGGGGSSILMQYIKANSKFIGIPMGLPQSIALS
SLMVAQ</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="135">
<INSDSeq>
<INSDSeq_length>43</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1413">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1412">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1414">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1415">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1416">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1417">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1418">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1419">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>32nd amino acid is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1420">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>42nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1421">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>43rd amino acid is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXKXLAAFTIRAAXX</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="136">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1423">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1422">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1424">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1425">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>18th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1426">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>20th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1427">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1428">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>31st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRNVPPIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGIC</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="137">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1430">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1429">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1431">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1432">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1433">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1434">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1435">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGIRNVP
PIFNDVYWIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="138">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1437">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1436">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1438">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1439">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1440">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1441">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1442">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGICRFR
GLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="139">
<INSDSeq>
<INSDSeq_length>65</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1444">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1443">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1445">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1446">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1447">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1448">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1449">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGIKTTK
QSFDLSVKAQYKKNKH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="140">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1451">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1450">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1452">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1453">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1454">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1455">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1456">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLSXXKXVAAWTLKAAXXILMQYIKANSKFIGIRNVP
PIFNDVYWIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="141">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1458">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1457">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1459">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1460">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1461">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1462">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1463">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLSXXKXVAAWTLKAAXXILMQYIKANSKFIGICRFR
GLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="142">
<INSDSeq>
<INSDSeq_length>65</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1465">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1464">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1466">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1467">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1468">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1469">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>29th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1470">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRFRGLISLSQVYLSXXKXVAAWTLKAAXXILMQYIKANSKFIGIKTTK
QSFDLSVKAQYKKNKH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="143">
<INSDSeq>
<INSDSeq_length>65</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1472">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1471">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1473">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>21st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1474">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>22nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1475">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1476">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1477">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>35th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXILMQYIKANSKFIG
IRNVPPIFNDVYWIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="144">
<INSDSeq>
<INSDSeq_length>65</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1479">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1478">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1480">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>21st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1481">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>22nd aminoacid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1482">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa)
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1483">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1484">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>35th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXILMQYIKANSKFIG
ICRFRGLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="145">
<INSDSeq>
<INSDSeq_length>70</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..70</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1486">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1485">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1487">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>21st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1488">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>22nd amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1489">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1490">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>34th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1491">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>35th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXILMQYIKANSKFIG
IKTTKQSFDLSVKAQYKKNKH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="146">
<INSDSeq>
<INSDSeq_length>80</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..80</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1493">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1492">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1494">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>36th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1495">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>37th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1496">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>39th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1497">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>49th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1498">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>50th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFXXKXVAAWTLKAAX
XILMQYIKANSKFIGIRNVPPIFNDVYWIAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="147">
<INSDSeq>
<INSDSeq_length>140</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..140</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1500">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1499">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFGGGGSGGGGGGSSI
LMQYIKANSKFIGIPMGLPQSIALSSLMVAQILMQYIKANSKFIGIPMGLPQSIALSSLMVAQGGGGSGG
GGGGSSCRFRGLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="148">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1502">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1501">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1503">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>13rd amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1504">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>24th amino acid(Xaa)is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PIFNDVYWIAFKXVAAWTLKAAKXVAAWTLKAACRFRGLISLSQ</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="149">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1506">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1505">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1507">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>11st amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1508">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>22nd amino acid(Xaa)is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PPIFNDVYWKXVAAWTLKAAKXVAAWTLKAARGLISLSQV</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="150">
<INSDSeq>
<INSDSeq_length>62</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..62</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1510">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide
(cyclic)</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1509">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1511">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1512">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>18th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1513">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>20th amino acid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1514">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>30th amino acid(Xaa) is a D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1515">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>31st amino acid(Xaa) is a 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CRNVPPIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGIRNV
PPIFNDVYWIAFC</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="151">
<INSDSeq>
<INSDSeq_length>80</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..80</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1517">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Concatamer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1516">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1518">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 36th, 50th, 66th, and 80th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1519">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 36th, 50th, 66th, and 80th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>66</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1520">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 36th, 50th, 66th, and 80th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>80</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1521">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 36th, 50th, 66th, and 80th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1522">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 37th, 49th, 67th, and 79th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1523">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 37th, 49th, 67th, and 79th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>67</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1524">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 37th, 49th, 67th, and 79th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>79</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1525">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 37th, 49th, 67th, and 79th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1526">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 39th and 69th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>69</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1527">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 39th and 69th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRGSHHHHHHGSDDDDKIVDRNVPPIFNDVYWIAFXXKXVAAWTLKAAX
XRNVPPIFNDVYWIAFXXKXVAAWTLKAAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="152">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1529">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Concatamer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1528">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1530">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 31st, 45th, 76th, and 90th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1531">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 31st, 45th, 76th, and 90th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>76</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1532">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 31st, 45th, 76th, and 90th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1533">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 31st, 45th, 76th, and 90th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1534">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 32nd, 44th, 77th, and 89th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1535">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 32nd, 44th, 77th, and 89th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>77</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1536">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 32nd, 44th, 77th, and 89th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>89</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1537">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 32nd, 44th, 77th, and 89th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1538">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 34th and 79th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>79</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1539">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 34th and 79th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFCRFRGLISLSQVYLSXXKXVAAWTLKAAXXRNVP
PIFNDVYWIAFCRFRGLISLSQVYLSXXKXVAAWTLKAAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="153">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1541">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Concatamer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1540">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1542">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1543">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>61</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1544">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>75</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1545">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1546">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd, and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1547">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd, and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>62</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1548">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd, and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>74</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1549">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd, and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1550">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 19th and 64th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>64</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1551">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 19th and 64th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXCRFRGLISLSQVYLSRNVP
PIFNDVYWIAFXXKXVAAWTLKAAXXCRFRGLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="154">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1553">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Concatamer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1552">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1554">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1555">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>61</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1556">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>75</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1557">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1558">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1559">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>62</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1560">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>74</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1561">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1562">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 19th and 64th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>64</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1563">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 19th and 64th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGIRNVP
PIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="155">
<INSDSeq>
<INSDSeq_length>120</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..120</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1565">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Concatamer</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1564">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1566">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 76th, and 90th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1567">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 76th, and 90th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>76</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1568">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 76th, and 90th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1569">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 76th, and 90th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1570">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 77th, and 89th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1571">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 77th, and 89th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1572">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 77th, and 89th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>77</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1573">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 77th, and 89th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>89</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1574">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 77th, and 89th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1575">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 19th and 79th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>79</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1576">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 19th and 79th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGIRNVP
PIFNDVYWIAFRNVPPIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGIRNVPPIFNDVYWIA
F</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="156">
<INSDSeq>
<INSDSeq_length>75</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..75</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1578">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>String-of-Beads</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1577">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1579">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1580">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>61</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1581">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>75</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1582">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 16th, 30th, 61st, and 75th amino
acid(Xaa) is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1583">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd, and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1584">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd, and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>62</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1585">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd, and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>74</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1586">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 17th, 29th, 62nd, and 74th
aminoacid(Xaa) is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1587">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 19th and 64th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>64</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1588">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Each of 19th and 64th aminoacid(Xaa) is
L-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXRNVPPIFNDVYWIAFCRFR
GLISLSQVYLSXXKXVAAWTLKAAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="157">
<INSDSeq>
<INSDSeq_length>152</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..152</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1590">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>String-of-Beads</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1589">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1591">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th, 30th, 46th, 62nd, 76th, 92nd,
108th, and 122nd Xaa is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1592">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th, 30th, 46th, 62nd, 76th, 92nd,
108th, and 122nd Xaa is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1593">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th, 30th, 46th, 62nd, 76th, 92nd,
108th, and 122nd Xaa is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>62</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1594">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th, 30th, 46th, 62nd, 76th, 92nd,
108th, and 122nd Xaa is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>76</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1595">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th, 30th, 46th, 62nd, 76th, 92nd,
108th, and 122nd Xaa is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>92</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1596">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th, 30th, 46th, 62nd, 76th, 92nd,
108th, and 122nd Xaa is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1597">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th, 30th, 46th, 62nd, 76th, 92nd,
108th, and 122nd Xaa is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>122</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1598">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>16th, 30th, 46th, 62nd, 76th, 92nd,
108th, and 122nd Xaa is a 6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1599">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th, 29th, 63rd, 75th, 109th, and 121st
Xaa is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1600">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th, 29th, 63rd, 75th, 109th, and 121st
Xaa is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>63</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1601">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th, 29th, 63rd, 75th, 109th, and 121st
Xaa is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>75</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1602">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th, 29th, 63rd, 75th, 109th, and 121st
Xaa is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>109</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1603">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th, 29th, 63rd, 75th, 109th, and 121st
Xaa is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>121</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1604">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>17th, 29th, 63rd, 75th, 109th, and 121st
Xaa is a D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1605">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th, 65th, and 111st Xaa
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>65</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1606">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th, 65th, and 111st Xaa
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1607">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>19th, 65th, and 111st Xaa
isL-cyclohexylalanins.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXRNVPPIFNDVYWIAFXRNV
PPIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGIXRNVPPIFNDVYWIAFXXKXVAAWTLKA
AXXILMQYIKANSKFIGICRFRGLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="158">
<INSDSeq>
<INSDSeq_length>272</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..272</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1609">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>String-of-Beads</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1608">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1610">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1611">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>61</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1612">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>75</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1613">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>106</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1614">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>120</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1615">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>136</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1616">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>150</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1617">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>166</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1618">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>182</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1619">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>196</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1620">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>212</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1621">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>228</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1622">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>242</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1623">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1624">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1625">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>62</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1626">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>74</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1627">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>107</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1628">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>119</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1629">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>137</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1630">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>149</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1631">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>183</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1632">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>195</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1633">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>229</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1634">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>241</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1635">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>D-form alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1636">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>L-cyclohexylalanins</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>64</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1637">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>L-cyclohexylalanins</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>109</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1638">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>L-cyclohexylalanins</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>139</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1639">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>L-cyclohexylalanins</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>185</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1640">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>L-cyclohexylalanins</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>231</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1641">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>L-cyclohexylalanins</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXRNVPPIFNDVYWIAFCRFR
GLISLSQVYLSXXKXVAAWTLKAAXXRNVPPIFNDVYWIAFCRFRGLISLSQVYLSXXKXVAAWTLKAAX
XRNVPPIFNDVYWIAFXXKXVAAWTLKAAXXRNVPPIFNDVYWIAFXRNVPPIFNDVYWIAFXXKXVAAW
TLKAAXXILMQYIKANSKFIGIXRNVPPIFNDVYWIAFXXKXVAAWTLKAAXXILMQYIKANSKFIGICR
FRGLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="159">
<INSDSeq>
<INSDSeq_length>6</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1643">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Auxiliary part</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1642">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RRRRRR</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="160">
<INSDSeq>
<INSDSeq_length>43</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1645">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1644">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1646">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 19 is
L-cyclohexylalanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1647">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 16 is 6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1648">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 17 is D-form
alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAACRFRGLISLSQVYLS</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="161">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1650">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1649">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1651">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 16 is 6-aminohexanoic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1652">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 17 is D-form
alanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1653">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 19 is
L-cyclohexylalanine</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAACR</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="162">
<INSDSeq>
<INSDSeq_length>10</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1655">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1654">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PLGFFPDHQL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="163">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1657">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1656">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>WPEANQVGAGAFGPGF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="164">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1659">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1658">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MQWNSTALHQALQDP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="165">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1661">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1660">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MQWNSTTFHQTLQDPRVRGLYFPAGG</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="166">
<INSDSeq>
<INSDSeq_length>10</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1663">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1662">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FFLLTRILTI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="167">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1665">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1664">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FFLLTRILTIPQSLD</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="168">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1667">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1666">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TSLNFLGGTTVCLGQ</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="169">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1669">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1668">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSPTSNHSPTSCPPIC</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="170">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1671">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1670">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IIFLFILLLCLIFLLVLLD</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="171">
<INSDSeq>
<INSDSeq_length>14</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1673">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1672">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CTTPAQGNSMFPSC</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="172">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1675">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1674">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CTKPTDGN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="173">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1677">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1676">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>WASVRFSW</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="174">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1679">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1678">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLPIFFCLW</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="175">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1681">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1680">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MDIDPYKEFGATVELLSFLP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="176">
<INSDSeq>
<INSDSeq_length>10</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1683">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1682">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLPSDFFPSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="177">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1685">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1684">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RDLLDTASALYREALESPEH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="178">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1687">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1686">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PHHTALRQAILCWGELMTLA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="179">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1689">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1688">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GRETVIEYLVSFGVW</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="180">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1691">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1690">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>EYLVSFGVWIRTPPA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="181">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1693">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1692">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VSFGVWIRTPPAYRPPNAPI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="182">
<INSDSeq>
<INSDSeq_length>10</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1695">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1694">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TVVRRRGRSP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="183">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1697">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1696">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VGPLTVNEKRRLKLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="184">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1699">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1698">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RHYLHTLWKAGILYK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="185">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1701">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1700">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ESRLVVDFSQFSRGN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="186">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1703">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1702">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LQSLTNLLSSNLSWL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="187">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1705">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1704">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SSNLSWLSLDVSAAF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="188">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1707">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1706">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LHLYSHPIILGFRKI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="189">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1709">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1708">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KQCFRKLPVNRPIDW</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="190">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1711">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1710">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LCQVFADATPTGWGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="191">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1713">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1712">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AANWILRGTSFVYVP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="192">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1715">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1714">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>EIRLKVFVLGGCRHK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="193">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1717">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Auxiliary part</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1716">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1718">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 16 is 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1719">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 17 is D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GSHHHHHHGSDDDDKXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="194">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1721">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Auxiliary part</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1720">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1722">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 1 is D-form
alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1723">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 2 is 6-aminohexanoic
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXGSHHHHHHGSDDDDK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="195">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1725">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1724">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KFVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="196">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1727">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1726">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KYVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="197">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1729">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1728">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1730">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa can be any naturally occurring amino
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="198">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1732">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1731">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1733">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1734">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1735">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1736">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1737">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXCRFRGLISLSQVYLS</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="199">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1739">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1738">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1740">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1741">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1742">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1743">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1744">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXXGSHHHHHHGSDDDDK</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="200">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1746">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1745">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1747">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1748">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1749">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1750">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1751">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GSHHHHHHGSDDDDKXXKXVAAWTLKAAXXRNVPPIFNDVYWIAF</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="201">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1753">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1752">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1754">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1755">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1756">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1757">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1758">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXKXVAAWTLKAAXXCRFRGLISLSQVYL
S</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="202">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1760">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1759">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1761">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1762">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1763">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1764">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1765">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFCRFRGLISLSQVYLSXXKXVAAWTLKAAXX</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="203">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1767">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1766">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1768">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1769">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXPKYVKQNTLKLATXCRFRGLISLSQVYLS</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="204">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1771">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1770">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1772">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1773">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1774">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1775">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1776">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="205">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1778">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1777">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1779">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFKXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="206">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1781">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1780">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1782">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFKXVAAWTLKAAHHHHHH</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="207">
<INSDSeq>
<INSDSeq_length>43</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1784">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1783">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1785">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1786">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1787">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAACRFRGLISLSQVYLS</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="208">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1789">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1788">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1790">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1791">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1792">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKXVAAWTLKAACR</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="209">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1794">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1793">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1795">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1796">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1797">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1798">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKFVAAWTLKAAXXCRFRGLISLSQVYLS</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="210">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1800">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1799">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1801">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1802">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1803">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1804">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKFVAAWTLKAAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="211">
<INSDSeq>
<INSDSeq_length>43</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1806">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1805">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1807">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1808">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKFVAAWTLKAACRFRGLISLSQVYLS</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="212">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1810">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1809">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1811">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1812">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFXXKFVAAWTLKAACR</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="213">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1814">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1813">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFCTKPTDGN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="214">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1816">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1815">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFLLPIFFCLW</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="215">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1818">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1817">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFFLPSDFFPSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="216">
<INSDSeq>
<INSDSeq_length>55</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..55</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1820">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1819">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1821">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 22 and position 39 are
D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1822">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 22 and position 39 are
D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1823">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 21 and position 40 are
6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1824">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 21 and position 40 are
6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXWPEANQVGAGAFGPGFXXCRFRGLISL
SQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="217">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1826">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1825">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1827">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 22 and position 43 are
D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1828">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 22 and position 43 are
D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1829">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 21 and position 44 are
6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1830">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 21 and position 44 are
6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXMDIDPYKEFGATVELLSFLPXXCRFRG
LISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="218">
<INSDSeq>
<INSDSeq_length>71</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..71</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1832">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1831">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1833">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 22 and position 55 are
D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>55</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1834">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 22 and position 55 are
D-form alanine.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1835">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 21 and position 56 are
6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>56</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1836">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Xaa at position 21 and position 56 are
6-aminohexanoic acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KTTKQSFDLSVKAQYKKNKHXXILMQYIKANSKFIGIPMGLPQSIALSS
LMVAQXXCRFRGLISLSQVYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="219">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1838">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1837">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFILMQYIKANSKFIGIHHHHHH</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="220">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1840">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Unit peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1839">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RNVPPIFNDVYWIAFMDIDPYKEFGATVELLSFLPHHHHHH</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="221">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1842">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B-cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1841">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RFRGLISLSQVYLDP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="222">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1844">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>B-cell epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1843">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SVCGCPVGHHDVVGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="223">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1846">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1845">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DIEKKIAKMEKASSVFNVVNS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="224">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1848">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1847">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YSGPLKAEIAQRLEDV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="225">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1850">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1849">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1851">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVKANTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="226">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1853">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1852">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1854">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVKANTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="227">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1856">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1855">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1857">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVKAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="228">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1859">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1858">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1860">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVKAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="229">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1862">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1861">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1863">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVWANTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="230">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1865">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1864">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1866">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVWANTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="231">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1868">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1867">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1869">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVWAYTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="232">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1871">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1870">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1872">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVWAVTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="233">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1874">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1873">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1875">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVYAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="234">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1877">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1876">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1878">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVYAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="235">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1880">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1879">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1881">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RXVRANTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="236">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1883">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1882">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1884">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVKAHTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="237">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1886">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1885">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1887">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVKAHTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="238">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1889">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1888">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1890">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAANTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="239">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1892">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1891">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1893">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAANTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="240">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1895">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1894">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1896">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAAYTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="241">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1898">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1897">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1899">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAAYTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="242">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1901">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1900">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1902">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="243">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1904">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1903">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1905">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAAKTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="244">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1907">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1906">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1908">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAAHTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="245">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1910">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1909">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1911">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAAATLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="246">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1913">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1912">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1914">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVAAWTLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="247">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1916">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Th epitope</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1915">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>VARIANT</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1917">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>X can be any amino
acid.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KXVMAATLKAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="248">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1919">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1918">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagnnngtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="249">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1921">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1920">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagttcgtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="250">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1923">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1922">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagtatgtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="251">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1925">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1924">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagnnngtggcagcttggaccctgaaggcagcannnnnnggatcgcatcaccatcaccatcacggatc
cgatgatgatgacaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="252">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1927">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1926">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagttcgtggcagcttggaccctgaaggcagcannnnnnggatcgcatcaccatcaccatcacggatc
cgatgatgatgacaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="253">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1929">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1928">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagtatgtggcagcttggaccctgaaggcagcannnnnnggatcgcatcaccatcaccatcacggatc
cgatgatgatgacaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="254">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1931">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1930">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggatcgcatcaccatcaccatcacggatccgatgatgatgacaagnnnn
nnaagnnngtggcagcttggaccctgaaggcagcannnnnncgtaatgttcctcctatcttcaatgatgt
ttattggattgcattc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="255">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1933">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1932">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggatcgcatcaccatcaccatcacggatccgatgatgatgacaagnnnn
nnaagttcgtggcagcttggaccctgaaggcagcannnnnncgtaatgttcctcctatcttcaatgatgt
ttattggattgcattc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="256">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1935">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1934">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggatcgcatcaccatcaccatcacggatccgatgatgatgacaagnnnn
nnaagtatgtggcagcttggaccctgaaggcagcannnnnncgtaatgttcctcctatcttcaatgatgt
ttattggattgcattc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="257">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1937">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1936">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaacgacaaagcaatcatttgatttaagtgtaaaagctcagtatnnnn
nnaagnnngtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="258">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1939">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1938">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaacgacaaagcaatcatttgatttaagtgtaaaagctcagtatnnnn
nnaagttcgtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="259">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1941">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1940">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaacgacaaagcaatcatttgatttaagtgtaaaagctcagtatnnnn
nnaagtatgtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="260">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1943">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1942">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattctgcc
gtttccgtggactgatttccctgtcccaggtttatctgtccnnnnnnaagnnngtggcagcttggaccct
gaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="261">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1945">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1944">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattctgcc
gtttccgtggactgatttccctgtcccaggtttatctgtccnnnnnnaagttcgtggcagcttggaccct
gaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="262">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1947">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1946">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattctgcc
gtttccgtggactgatttccctgtcccaggtttatctgtccnnnnnnaagtatgtggcagcttggaccct
gaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="263">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1949">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1948">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnc
ctaagtatgtgaagcagaatacactgaagctggcaaccnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="264">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1951">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1950">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagnnngtggcagcttggaccctgaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="265">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1953">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1952">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagttcgtggcagcttggaccctgaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="266">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1955">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1954">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagtatgtggcagcttggaccctgaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="267">
<INSDSeq>
<INSDSeq_length>78</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..78</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1957">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1956">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagn
nngtggcagcttggaccctgaaggcagca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="268">
<INSDSeq>
<INSDSeq_length>78</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..78</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1959">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1958">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagt
tcgtggcagcttggaccctgaaggcagca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="269">
<INSDSeq>
<INSDSeq_length>78</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..78</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1961">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1960">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagt
atgtggcagcttggaccctgaaggcagca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="270">
<INSDSeq>
<INSDSeq_length>96</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..96</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1963">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1962">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagn
nngtggcagcttggaccctgaaggcagcacatcaccatcaccatcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="271">
<INSDSeq>
<INSDSeq_length>96</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..96</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1965">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1964">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagt
tcgtggcagcttggaccctgaaggcagcacatcaccatcaccatcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="272">
<INSDSeq>
<INSDSeq_length>96</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..96</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1967">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1966">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagt
atgtggcagcttggaccctgaaggcagcacatcaccatcaccatcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="273">
<INSDSeq>
<INSDSeq_length>129</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..129</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1969">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1968">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagnnngtggcagcttggaccctgaaggcagcatgccgtttccgtggactgatttccctgtcccaggt
ttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="274">
<INSDSeq>
<INSDSeq_length>129</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..129</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1971">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1970">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagttcgtggcagcttggaccctgaaggcagcatgccgtttccgtggactgatttccctgtcccaggt
ttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="275">
<INSDSeq>
<INSDSeq_length>129</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..129</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1973">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1972">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagtatgtggcagcttggaccctgaaggcagcatgccgtttccgtggactgatttccctgtcccaggt
ttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="276">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1975">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1974">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagnnngtggcagcttggaccctgaaggcagcatgccgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="277">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1977">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1976">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagttcgtggcagcttggaccctgaaggcagcatgccgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="278">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1979">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>DNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1978">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagtatgtggcagcttggaccctgaaggcagcatgccgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="279">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1981">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1980">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagnnngtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="280">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1983">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1982">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagttcgtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="281">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1985">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1984">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagtatgtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="282">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1987">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1986">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagnnngtggcagcttggaccctgaaggcagcannnnnnggatcgcatcaccatcaccatcacggatc
cgatgatgatgacaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="283">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1989">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1988">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagttcgtggcagcttggaccctgaaggcagcannnnnnggatcgcatcaccatcaccatcacggatc
cgatgatgatgacaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="284">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1991">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1990">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagtatgtggcagcttggaccctgaaggcagcannnnnnggatcgcatcaccatcaccatcacggatc
cgatgatgatgacaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="285">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1993">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1992">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggatcgcatcaccatcaccatcacggatccgatgatgatgacaagnnnn
nnaagnnngtggcagcttggaccctgaaggcagcannnnnncgtaatgttcctcctatcttcaatgatgt
ttattggattgcattc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="286">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1995">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1994">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggatcgcatcaccatcaccatcacggatccgatgatgatgacaagnnnn
nnaagttcgtggcagcttggaccctgaaggcagcannnnnncgtaatgttcctcctatcttcaatgatgt
ttattggattgcattc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="287">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1997">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1996">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggatcgcatcaccatcaccatcacggatccgatgatgatgacaagnnnn
nnaagtatgtggcagcttggaccctgaaggcagcannnnnncgtaatgttcctcctatcttcaatgatgt
ttattggattgcattc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="288">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1999">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1998">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaacgacaaagcaatcatttgatttaagtgtaaaagctcagtatnnnn
nnaagnnngtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="289">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2001">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2000">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaacgacaaagcaatcatttgatttaagtgtaaaagctcagtatnnnn
nnaagttcgtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="290">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2003">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2002">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaacgacaaagcaatcatttgatttaagtgtaaaagctcagtatnnnn
nnaagtatgtggcagcttggaccctgaaggcagcannnnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="291">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2005">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2004">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattctgcc
gtttccgtggactgatttccctgtcccaggtttatctgtccnnnnnnaagnnngtggcagcttggaccct
gaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="292">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2007">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2006">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattctgcc
gtttccgtggactgatttccctgtcccaggtttatctgtccnnnnnnaagttcgtggcagcttggaccct
gaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="293">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2009">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2008">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattctgcc
gtttccgtggactgatttccctgtcccaggtttatctgtccnnnnnnaagtatgtggcagcttggaccct
gaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="294">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2011">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2010">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnc
ctaagtatgtgaagcagaatacactgaagctggcaaccnnntgccgtttccgtggactgatttccctgtc
ccaggtttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="295">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2013">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2012">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagnnngtggcagcttggaccctgaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="296">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2015">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2014">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagttcgtggcagcttggaccctgaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="297">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2017">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2016">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagtatgtggcagcttggaccctgaaggcagcannnnnn</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="298">
<INSDSeq>
<INSDSeq_length>78</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..78</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2019">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2018">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagn
nngtggcagcttggaccctgaaggcagca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="299">
<INSDSeq>
<INSDSeq_length>78</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..78</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2021">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2020">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagt
tcgtggcagcttggaccctgaaggcagca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="300">
<INSDSeq>
<INSDSeq_length>78</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..78</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2023">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2022">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagt
atgtggcagcttggaccctgaaggcagca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="301">
<INSDSeq>
<INSDSeq_length>96</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..96</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2025">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2024">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagn
nngtggcagcttggaccctgaaggcagcacatcaccatcaccatcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="302">
<INSDSeq>
<INSDSeq_length>96</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..96</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2027">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2026">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagt
tcgtggcagcttggaccctgaaggcagcacatcaccatcaccatcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="303">
<INSDSeq>
<INSDSeq_length>96</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..96</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2029">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2028">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcaagt
atgtggcagcttggaccctgaaggcagcacatcaccatcaccatcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="304">
<INSDSeq>
<INSDSeq_length>129</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..129</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2031">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2030">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagnnngtggcagcttggaccctgaaggcagcatgccgtttccgtggactgatttccctgtcccaggt
ttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="305">
<INSDSeq>
<INSDSeq_length>129</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..129</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2033">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2032">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagttcgtggcagcttggaccctgaaggcagcatgccgtttccgtggactgatttccctgtcccaggt
ttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="306">
<INSDSeq>
<INSDSeq_length>129</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..129</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2035">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2034">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagtatgtggcagcttggaccctgaaggcagcatgccgtttccgtggactgatttccctgtcccaggt
ttatctgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="307">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2037">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2036">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagnnngtggcagcttggaccctgaaggcagcatgccgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="308">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2039">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2038">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagttcgtggcagcttggaccctgaaggcagcatgccgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="309">
<INSDSeq>
<INSDSeq_length>90</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..90</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2041">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>RNA encoding unit
peptide</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2040">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaatgttcctcctatcttcaatgatgtttattggattgcattcnnnn
nnaagtatgtggcagcttggaccctgaaggcagcatgccgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| АНТИГЕННЫЕ ПЕПТИДЫ ДЛЯ ПРОФИЛАКТИКИ И ЛЕЧЕНИЯ В-КЛЕТОЧНЫХ ГЕМОБЛАСТОЗОВ | 2020 |
|
RU2833806C1 |
| ПОЛИПЕПТИД, ВКЛЮЧАЮЩИЙ АНТИГЕНСВЯЗЫВАЮЩИЙ ДОМЕН И ТРАНСПОРТИРУЮЩИЙ СЕГМЕНТ | 2018 |
|
RU2841528C2 |
| Способ анализа соматических мутаций в генах IDH1 и IDH2 с использованием LNA-блокирующей ПЦР и гибридизации с биологическим микрочипом | 2024 |
|
RU2839291C1 |
| ХИМЕРНЫЕ ЦИТОКИНОВЫЕ РЕЦЕПТОРЫ, НЕСУЩИЕ ЭКТОДОМЕН PD-1 | 2020 |
|
RU2828214C2 |
| N-галлаты синтетических пептидов с антиоксидантной активностью | 2023 |
|
RU2828796C1 |
| БИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА И ИХ ПРИМЕНЕНИЯ | 2022 |
|
RU2821577C2 |
| АНТИТЕЛА ПРОТИВ ПРОПЕРДИНА И ИХ ПОЛУЧЕНИЕ | 2021 |
|
RU2830308C1 |
| ПЕПТИДЫ, ОБЛАДАЮЩИЕ АНТИБАКТЕРИАЛЬНОЙ АКТИВНОСТЬЮ | 2024 |
|
RU2837372C1 |
| АНТИСМЫСЛОВЫЕ ОЛИГОНУКЛЕОТИДЫ, НАЦЕЛЕННЫЕ НА ГЕН CAV3.1, И ИХ ПРИМЕНЕНИЯ | 2022 |
|
RU2839721C2 |
| Нуклеотидная последовательность, кодирующая фермент литиказу, и панель олигонуклеотидов для получения синтетической нуклеотидной последовательности гена литиказы | 2023 |
|
RU2826150C1 |
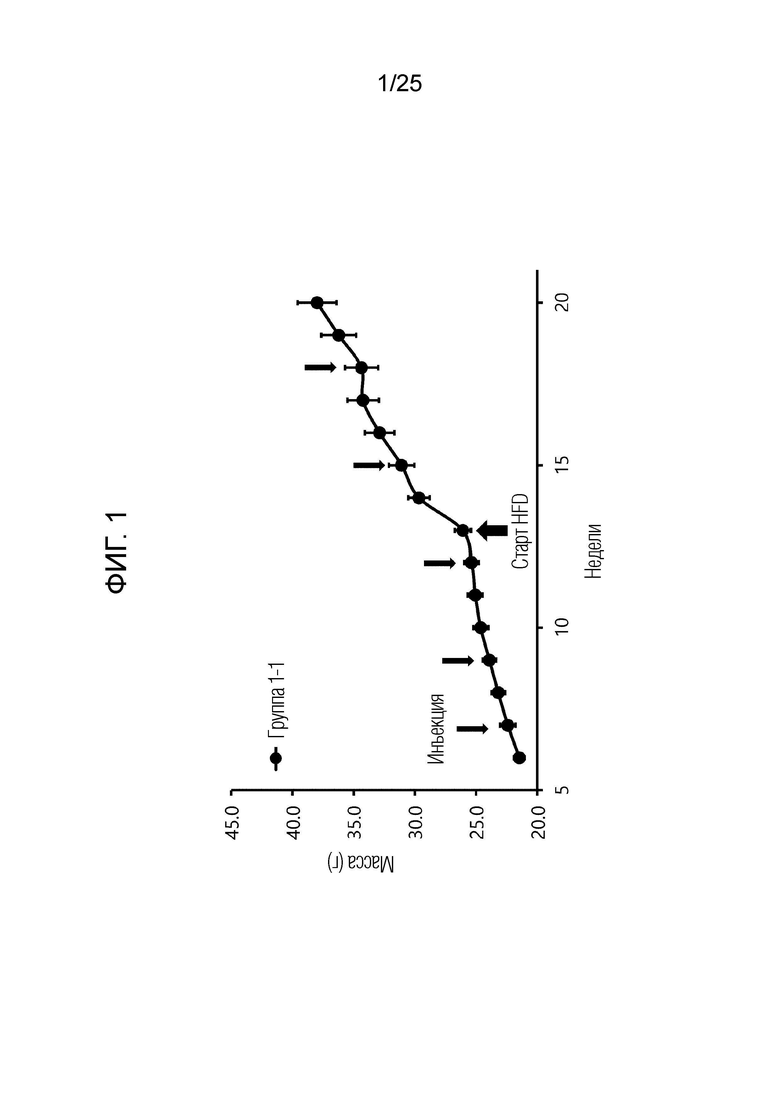
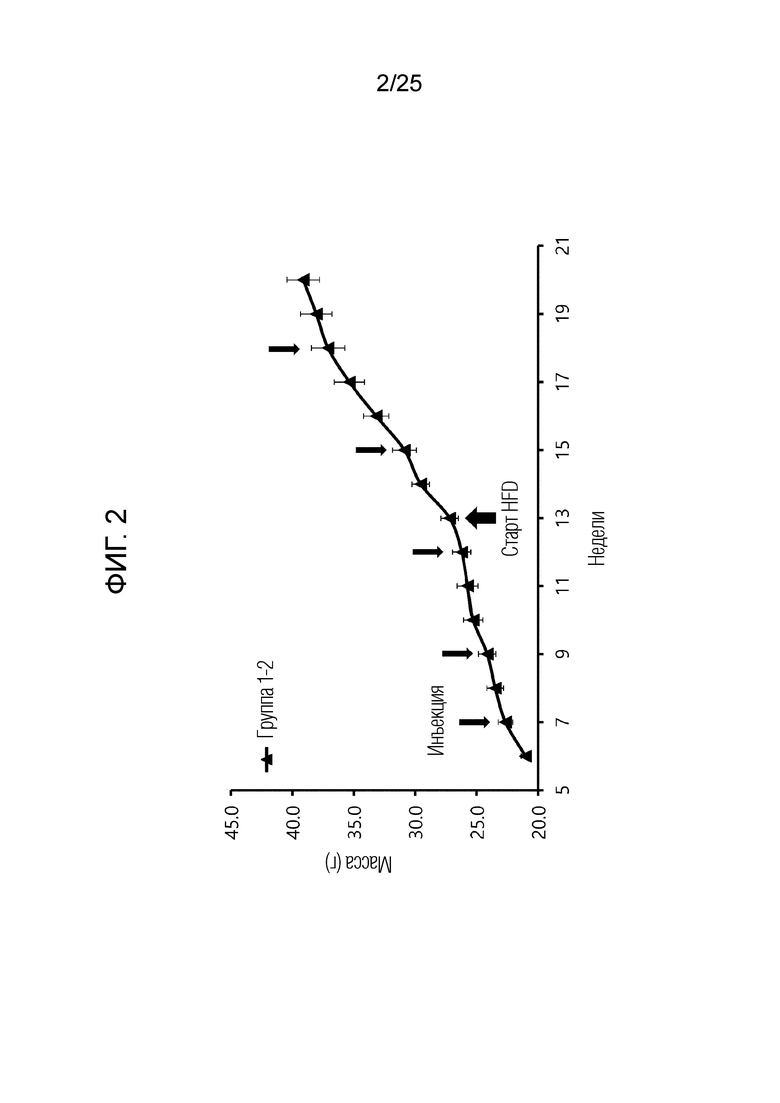
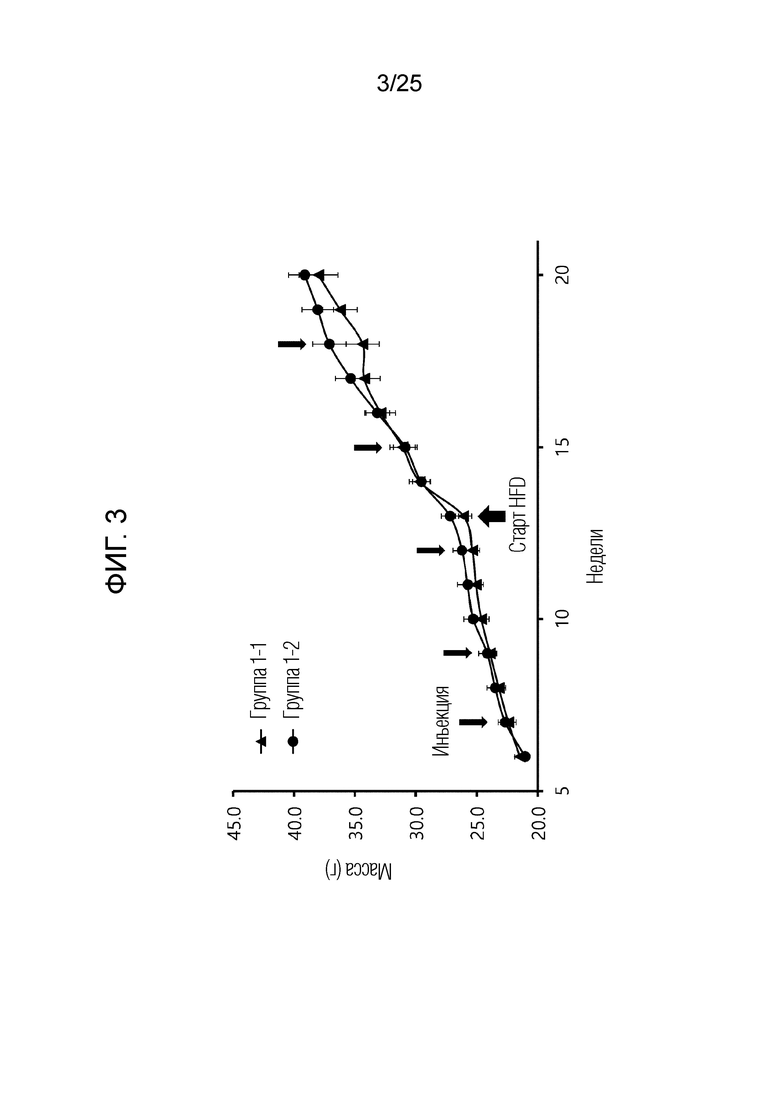
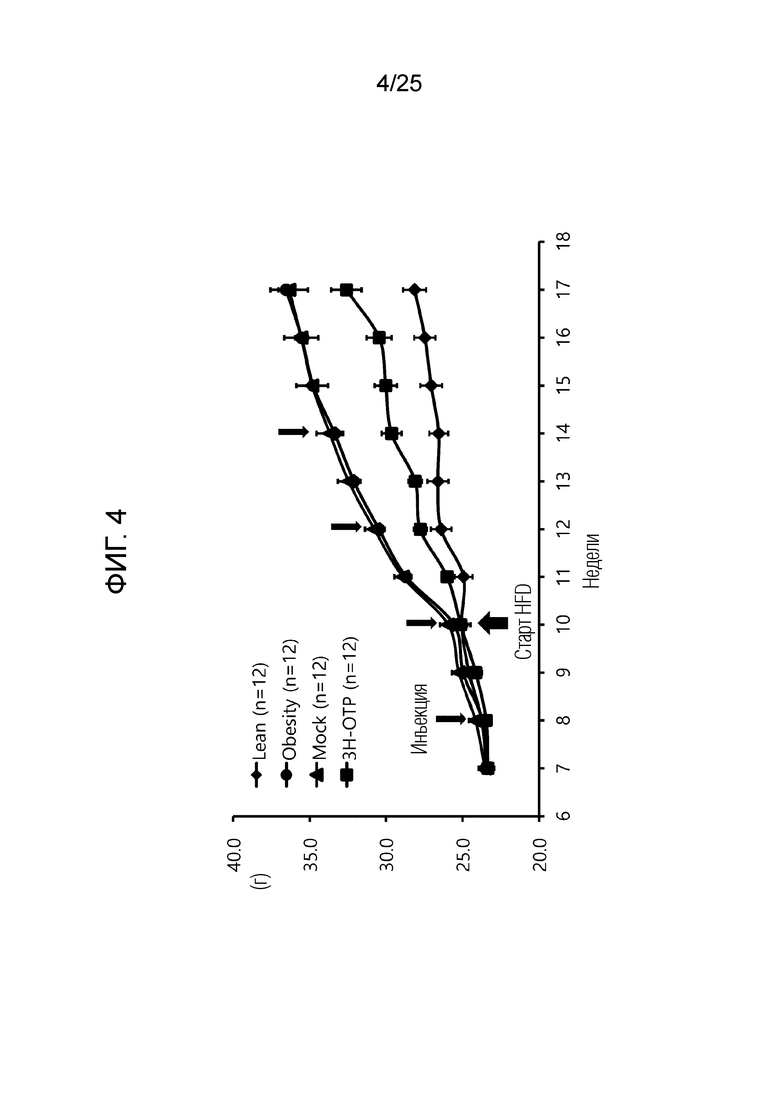
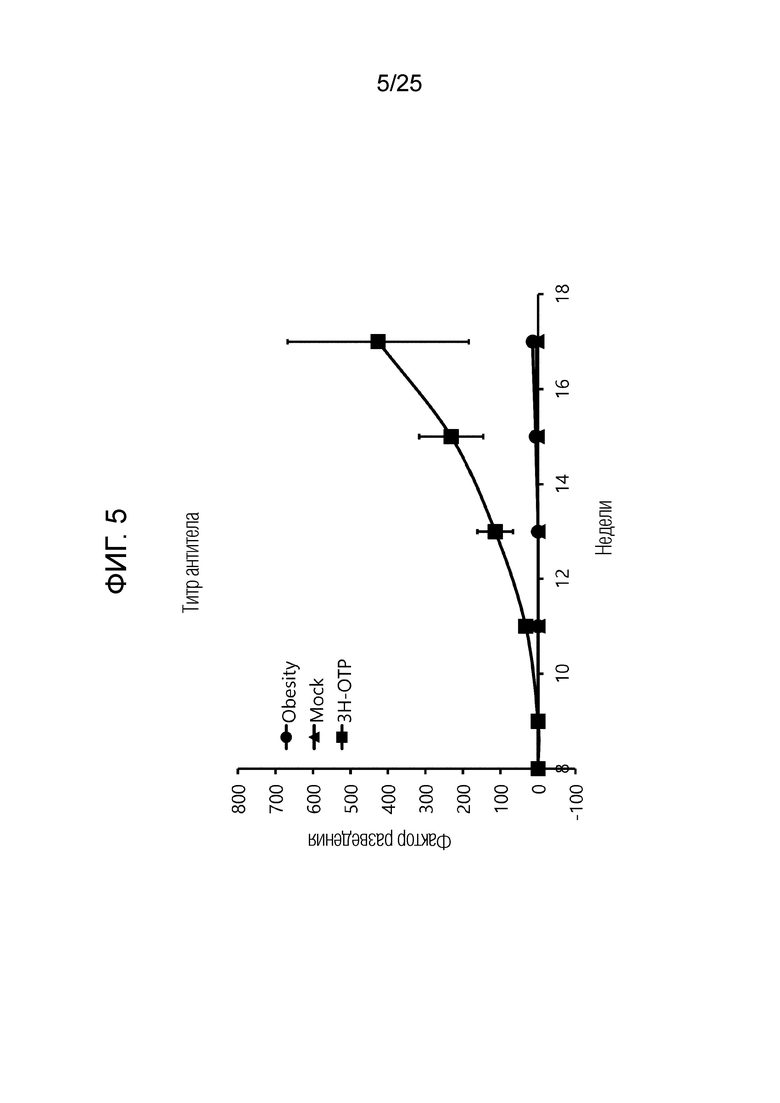
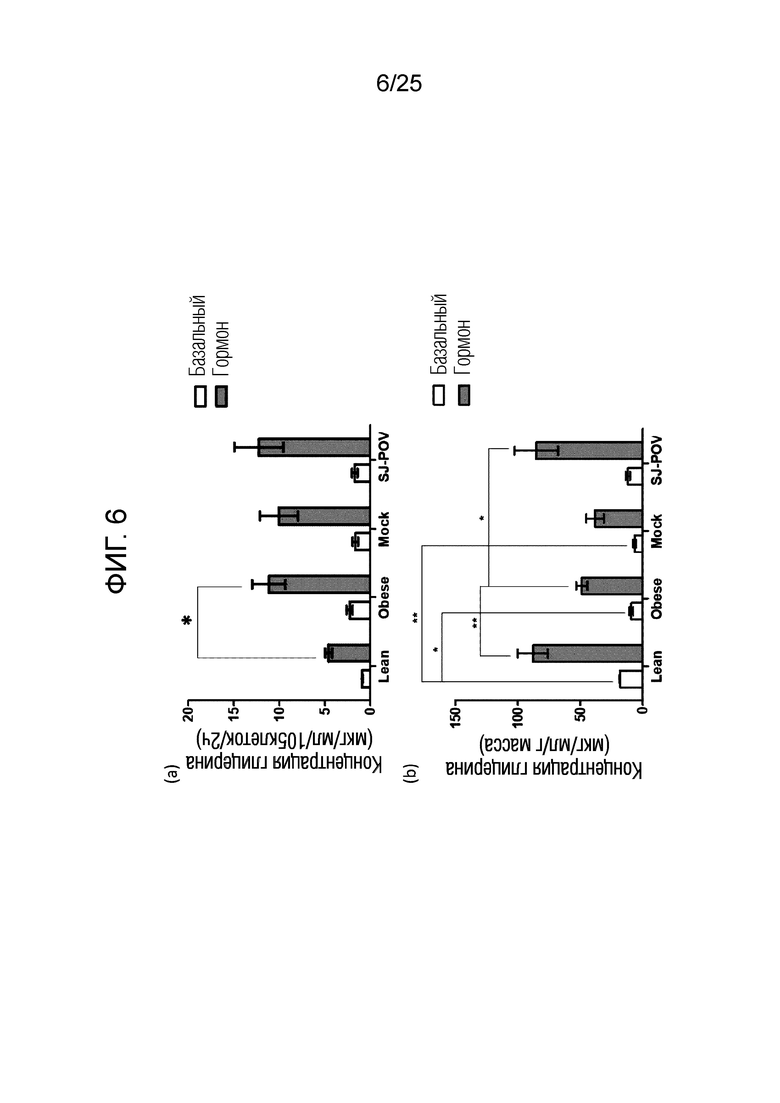


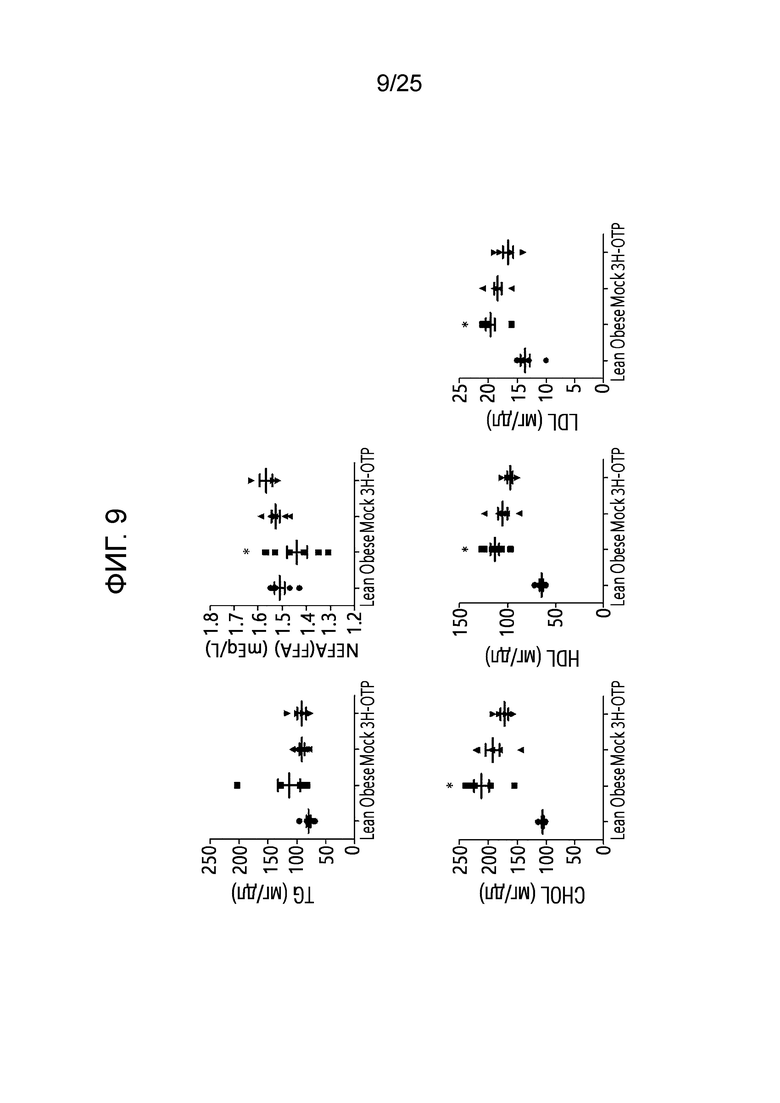
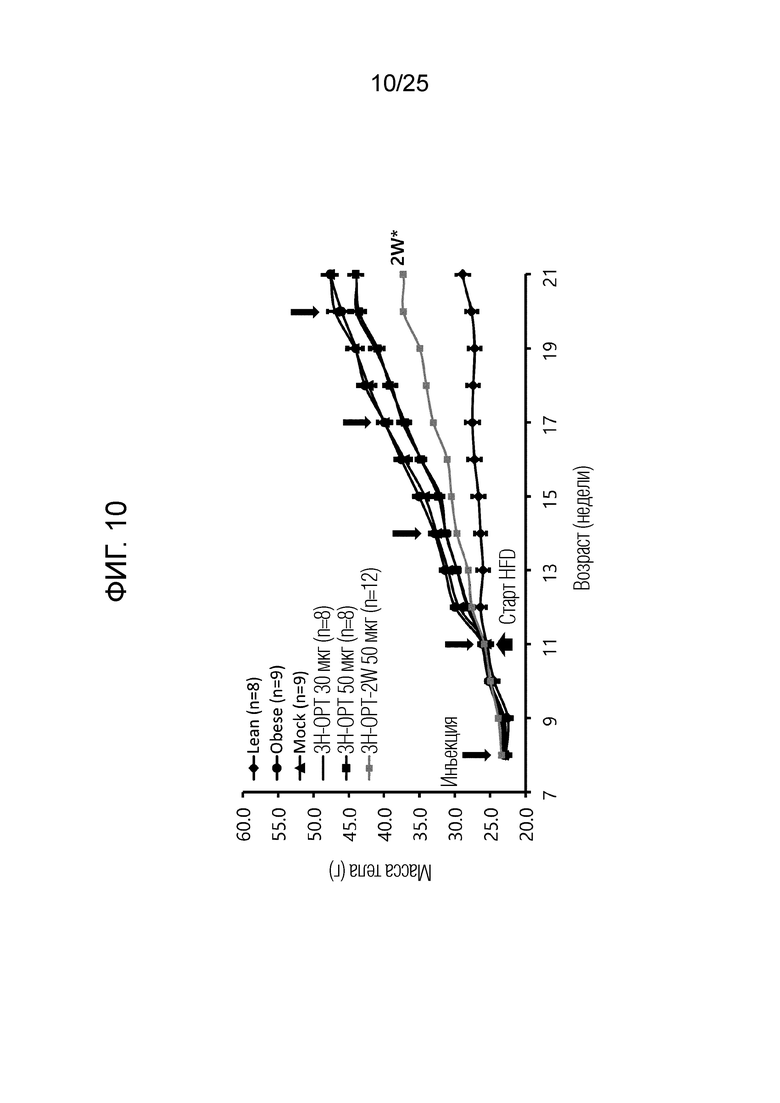
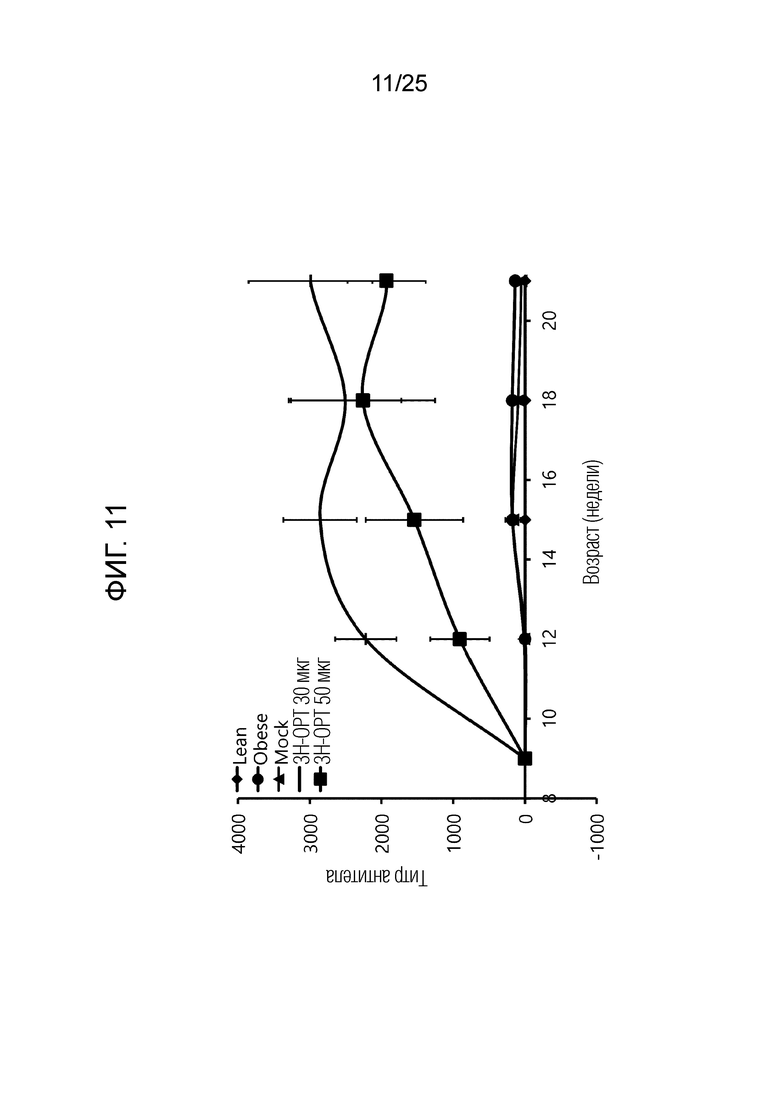
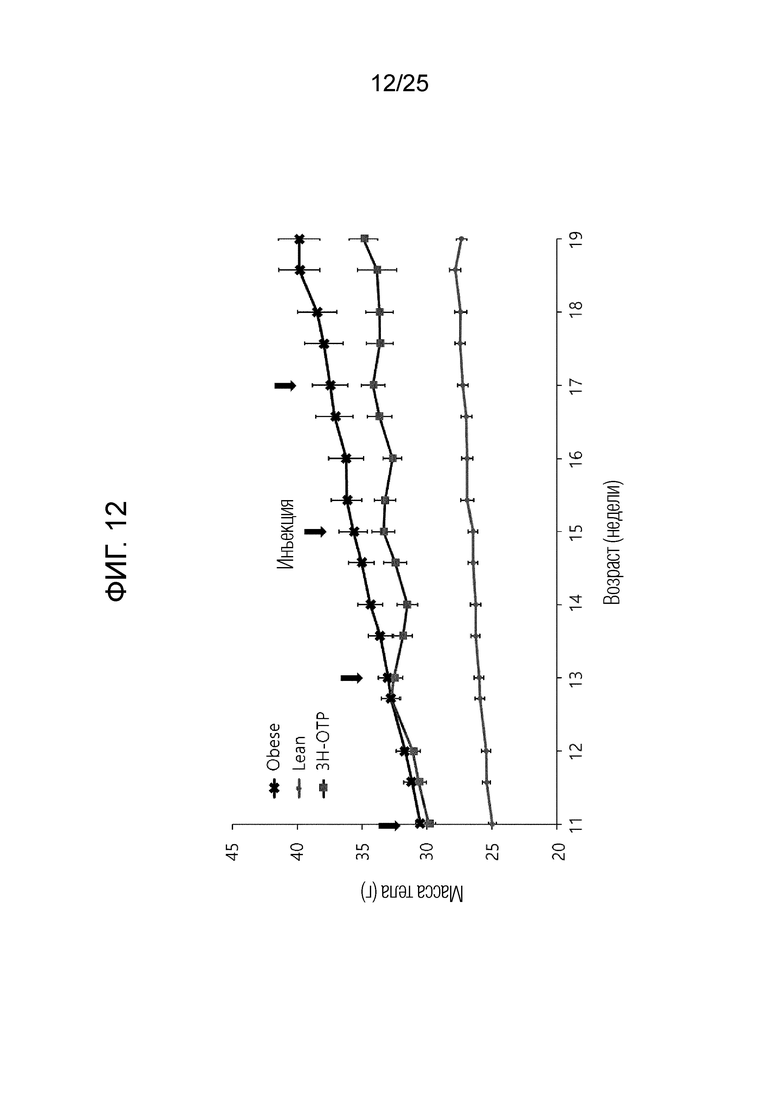
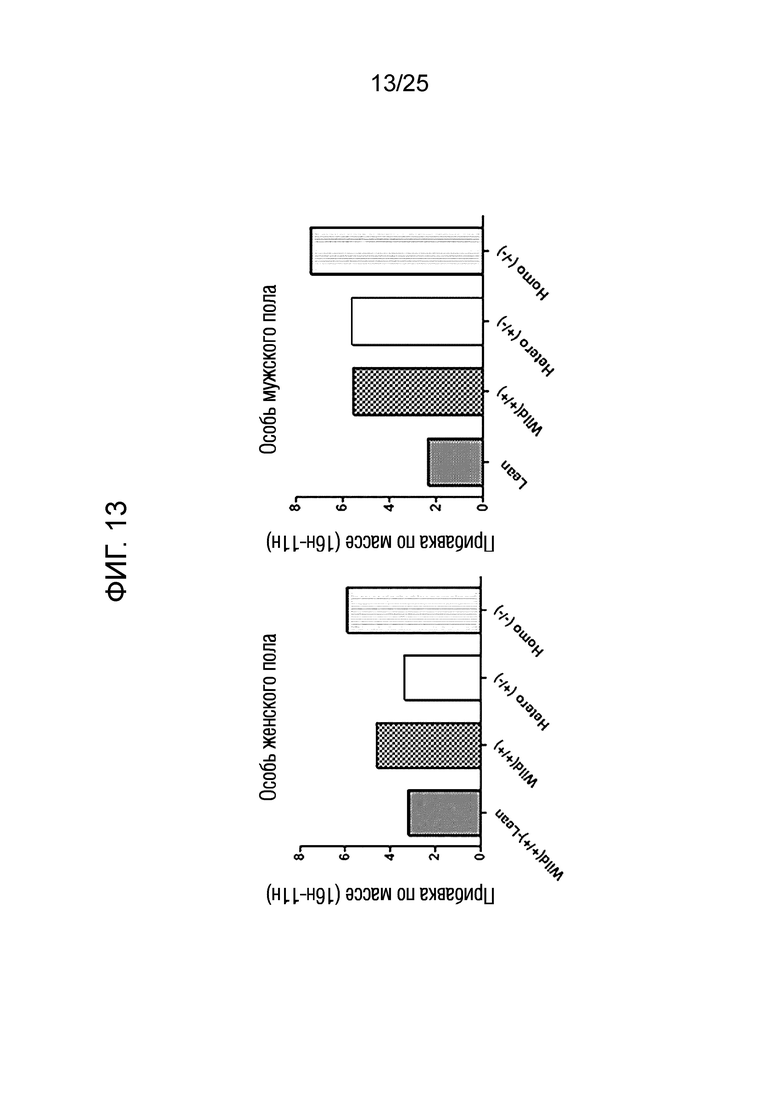
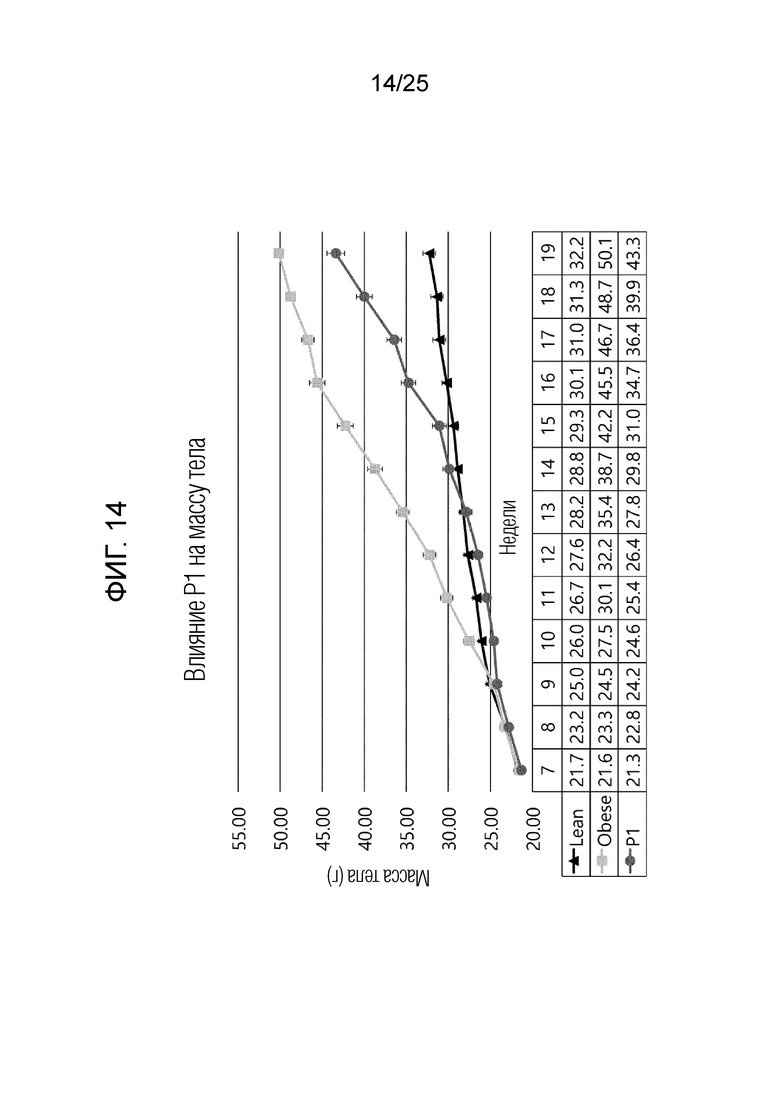
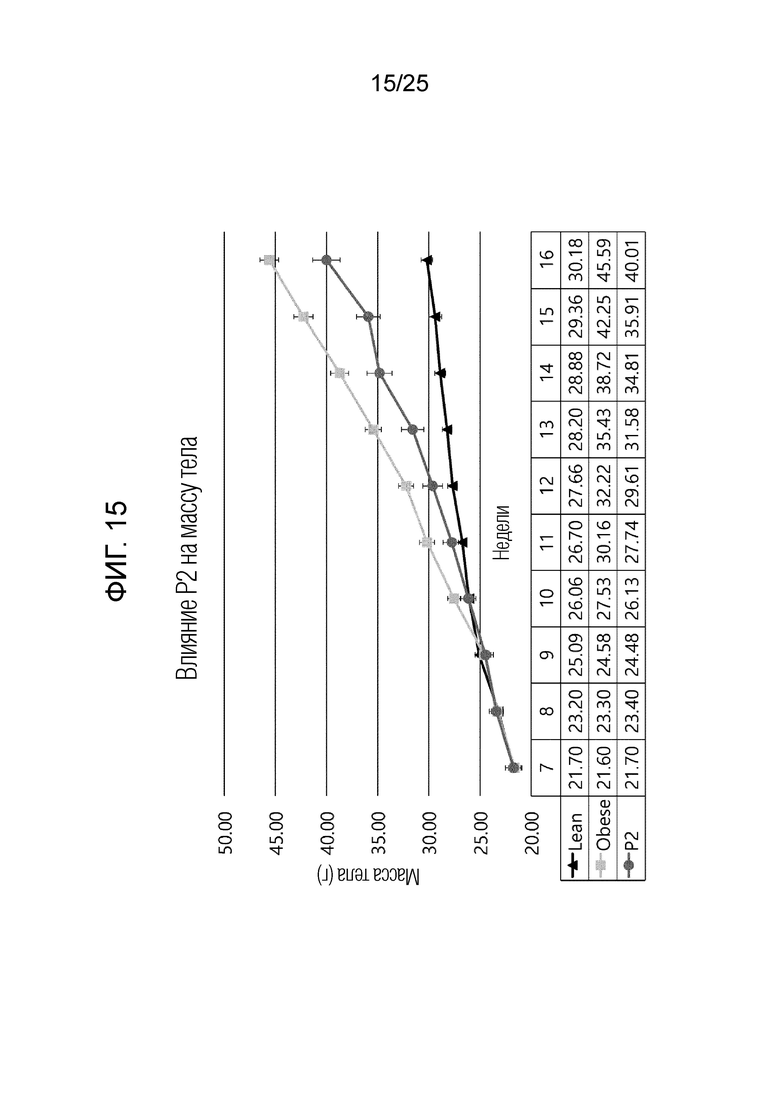
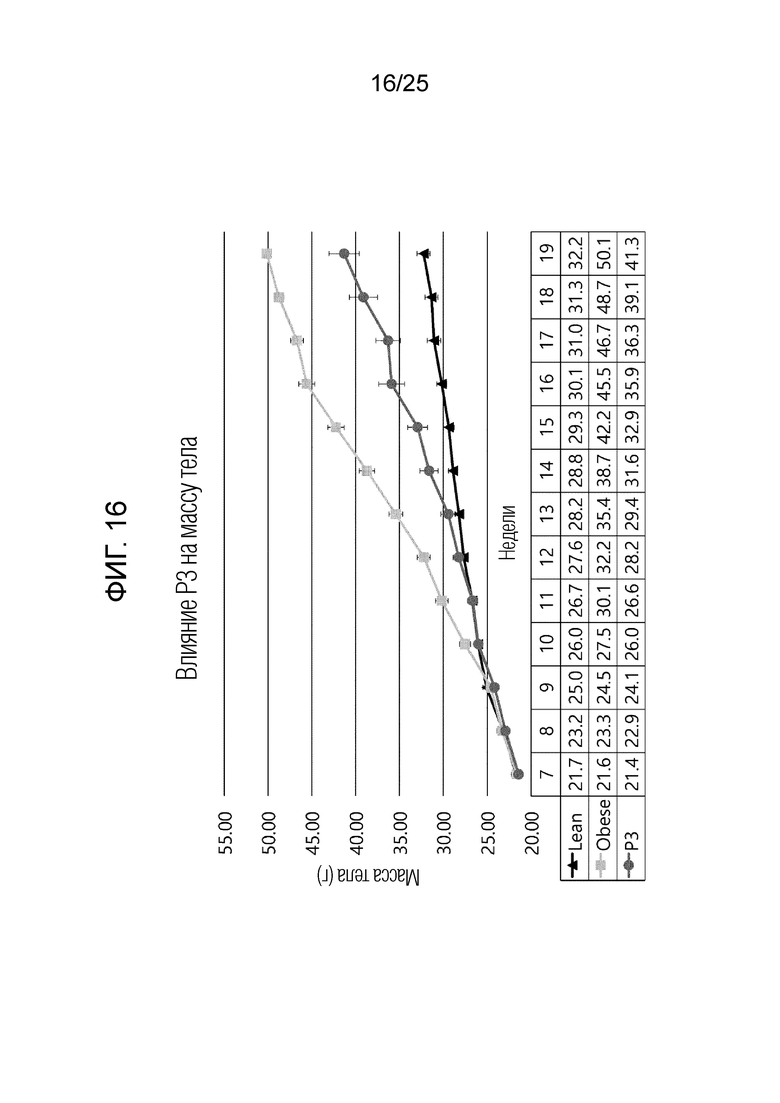
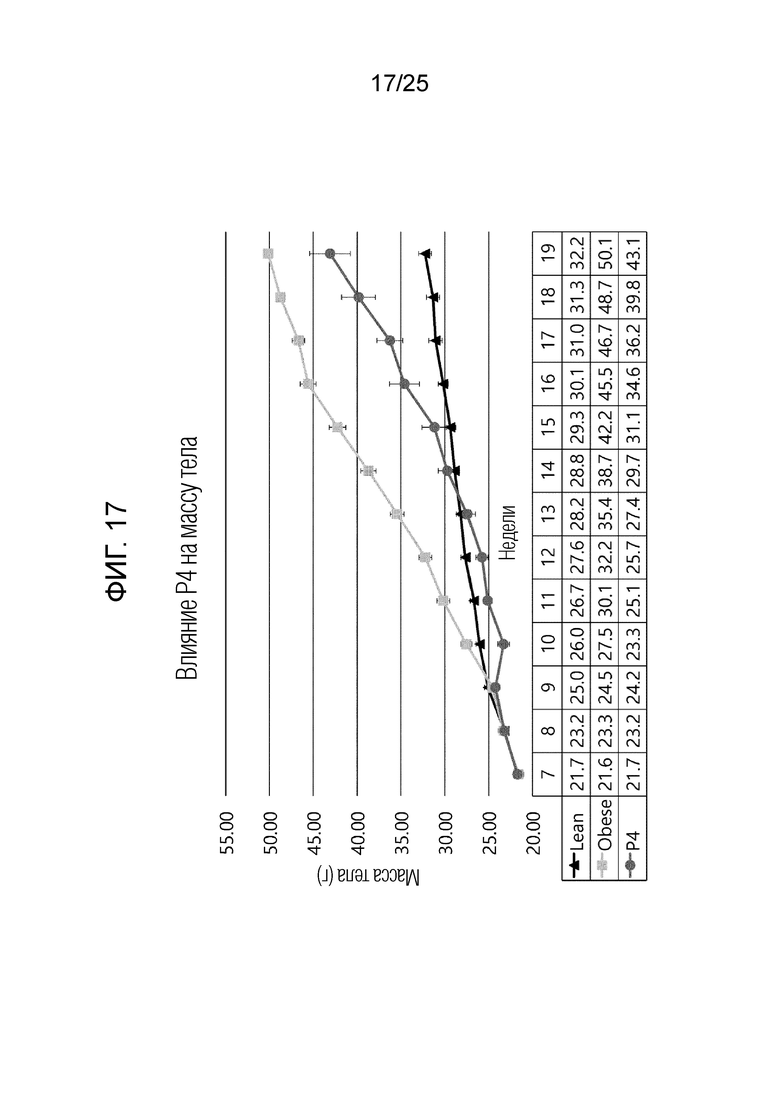
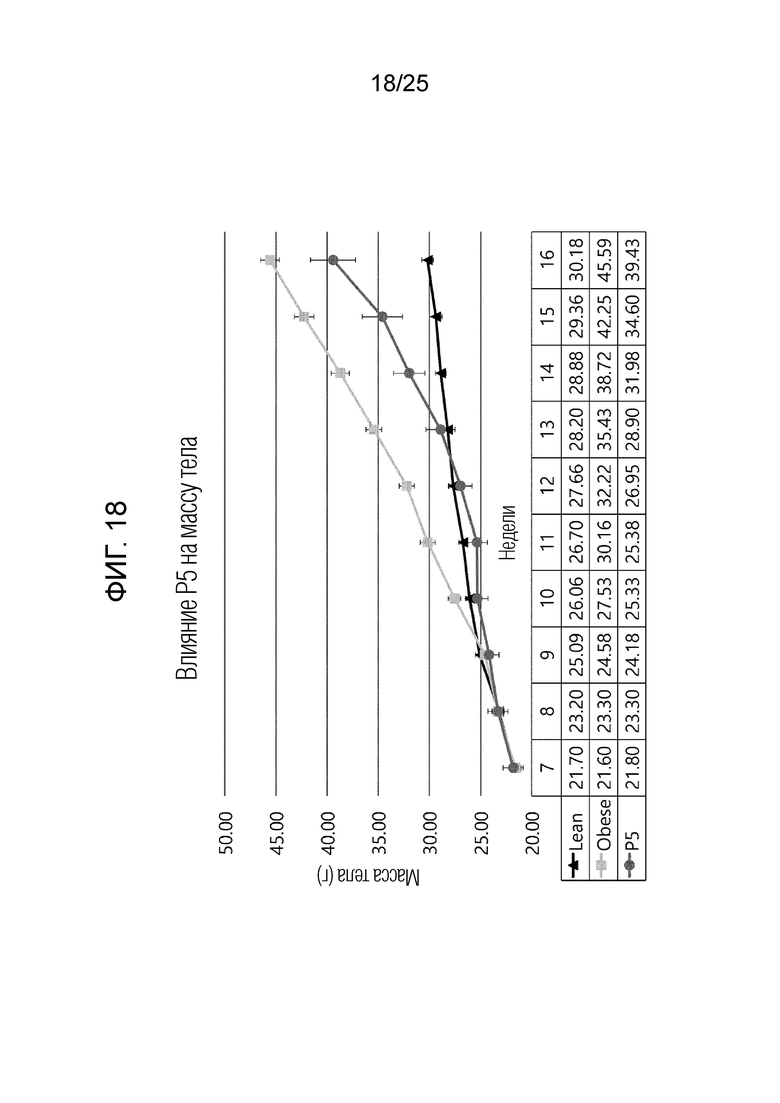
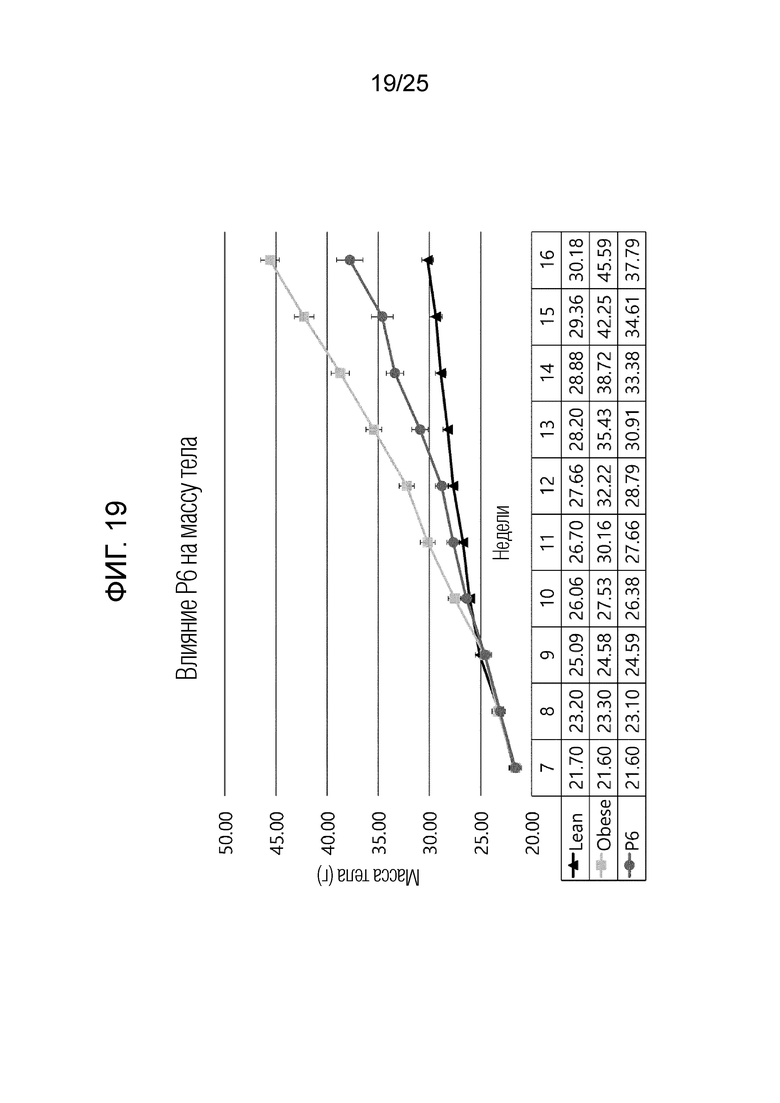
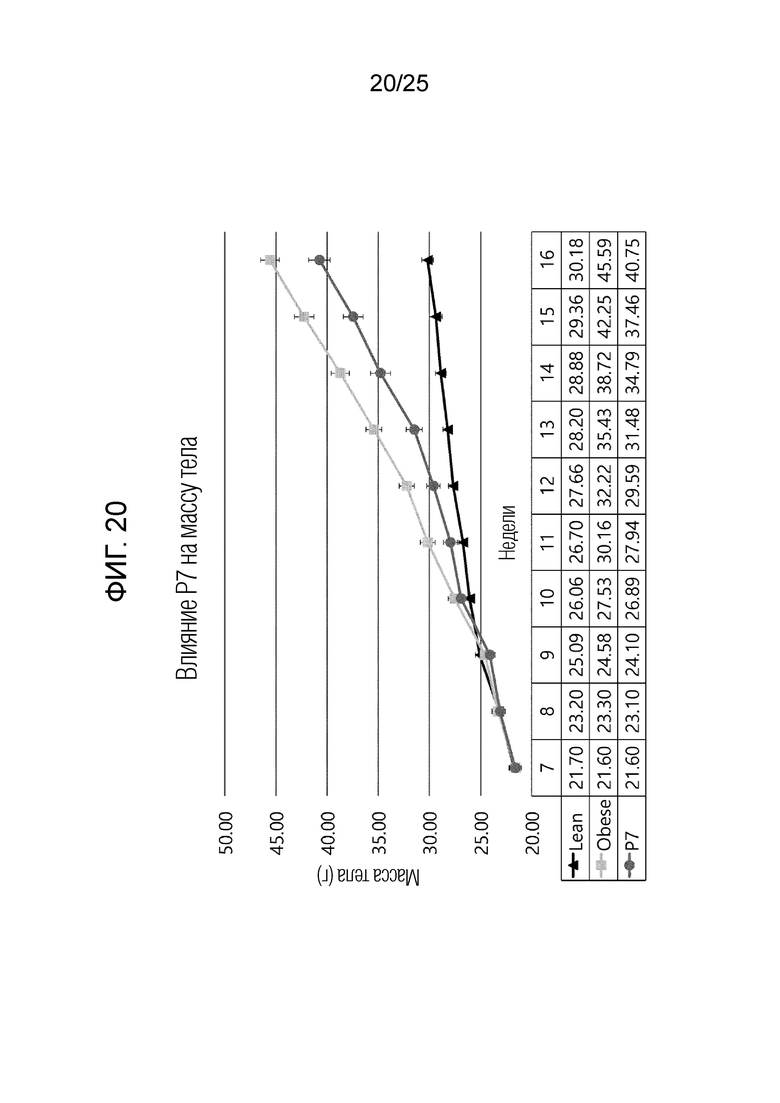
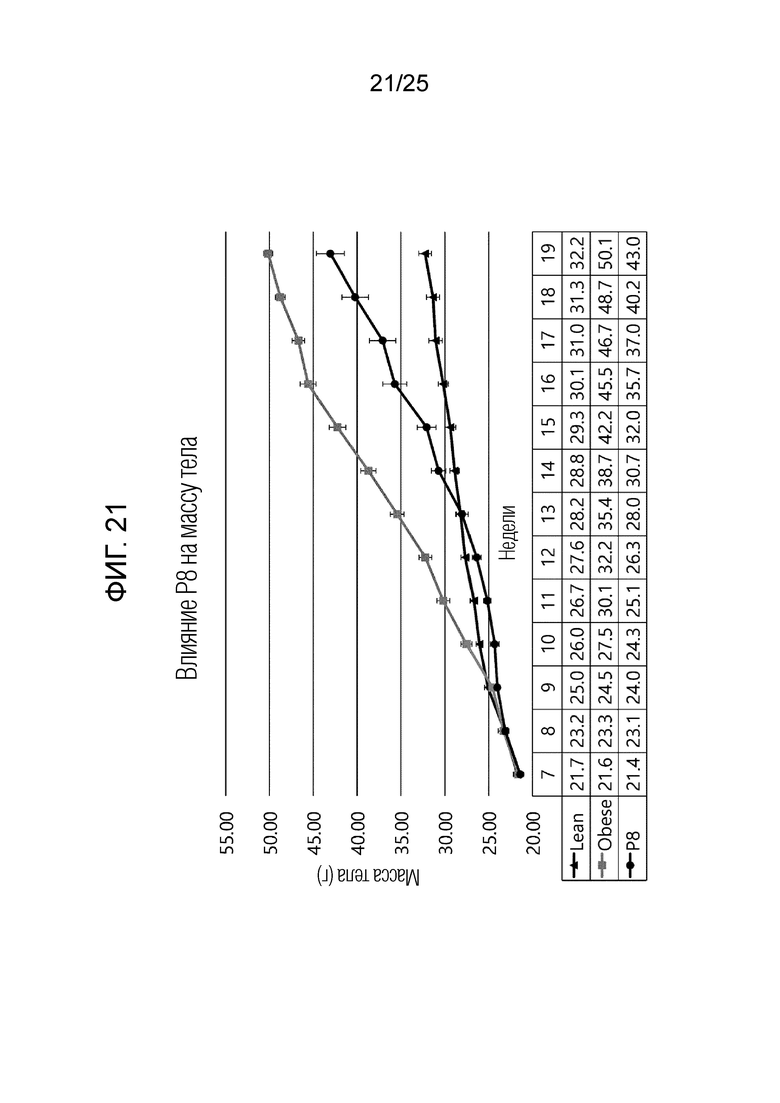
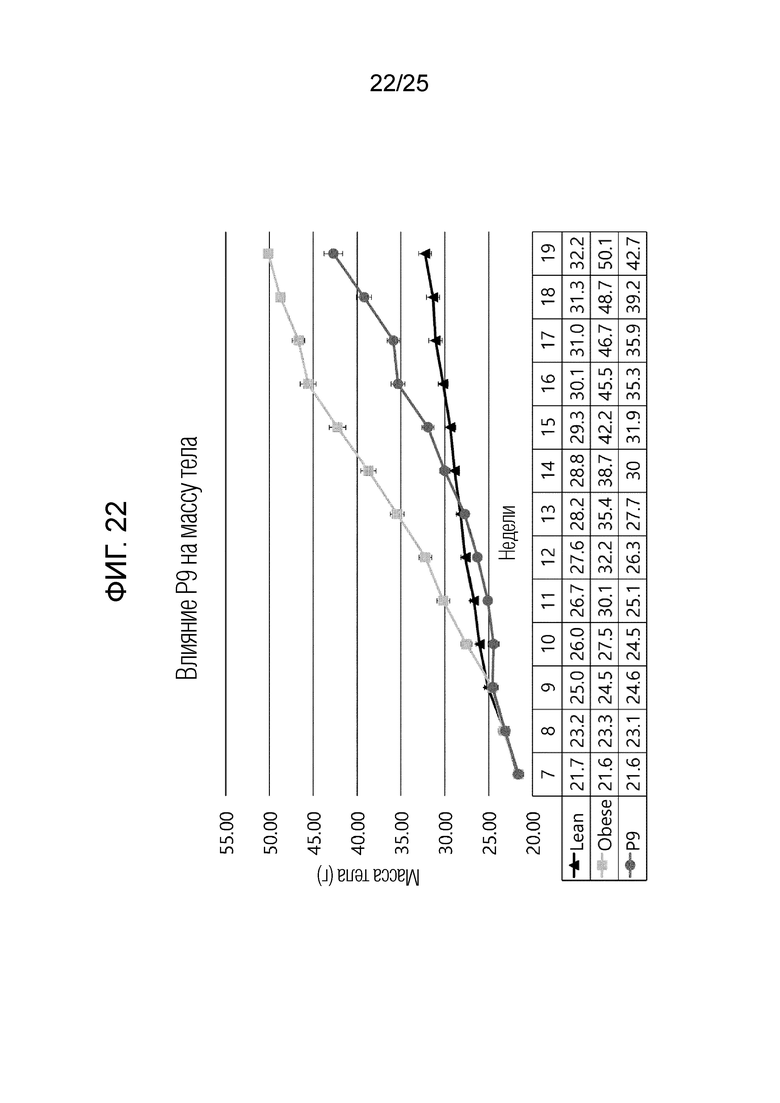
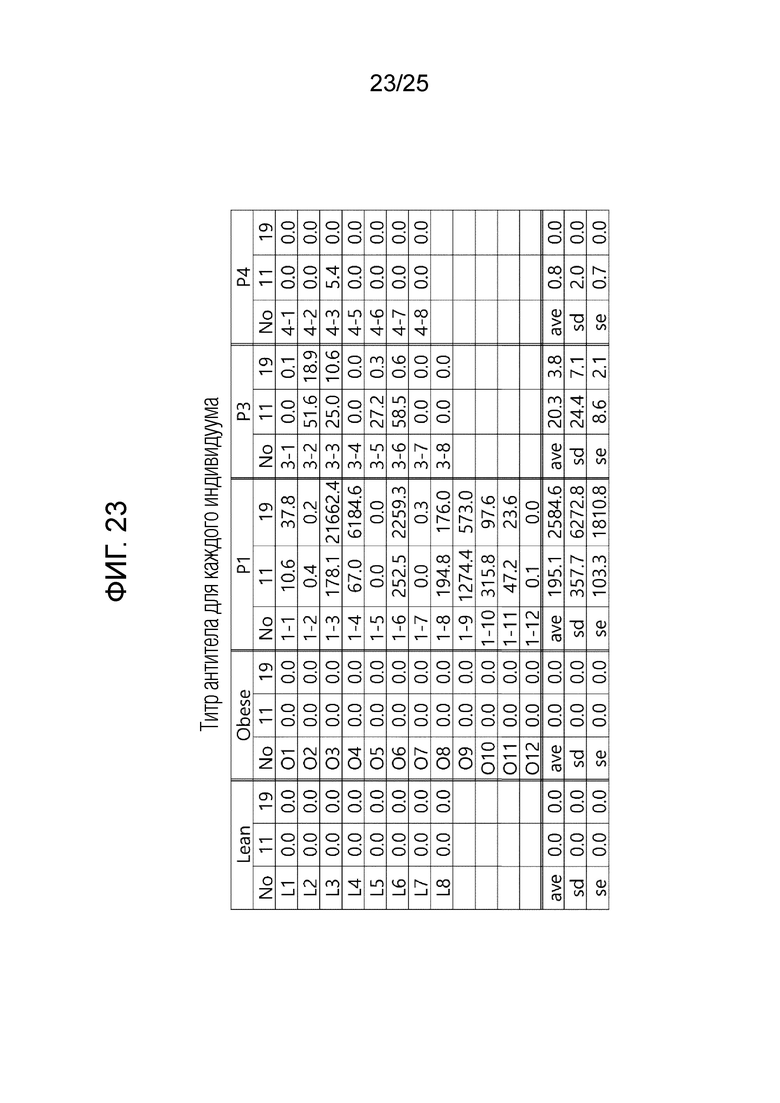
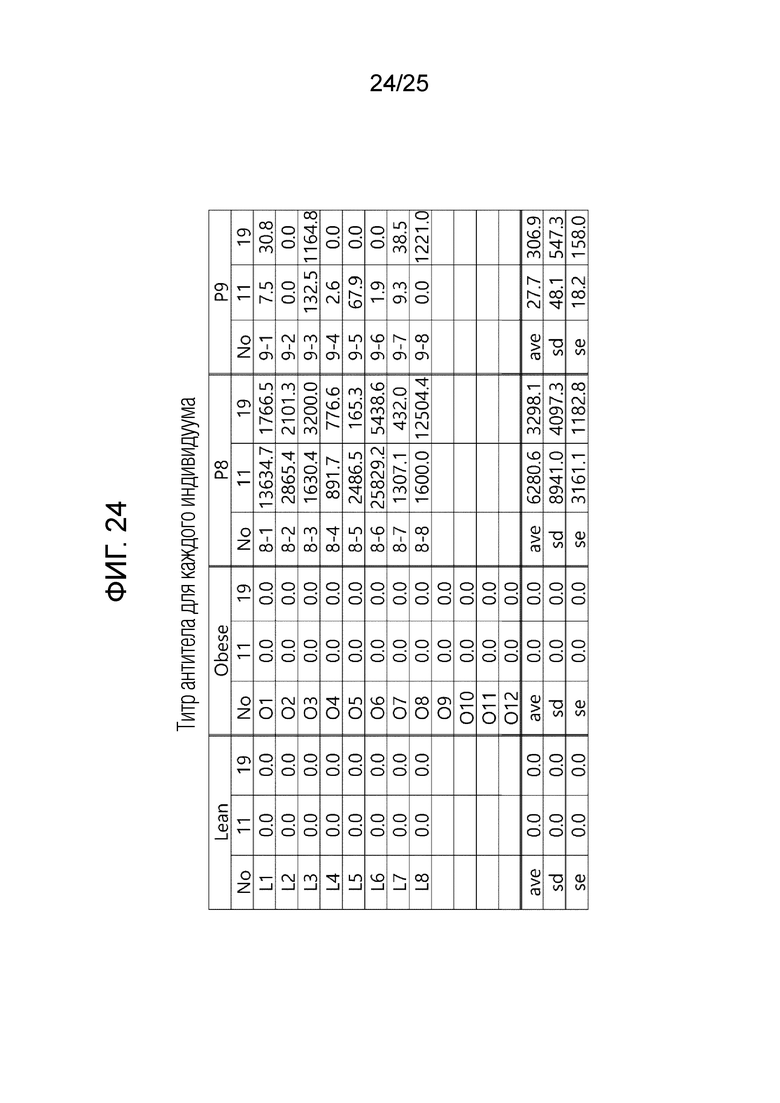
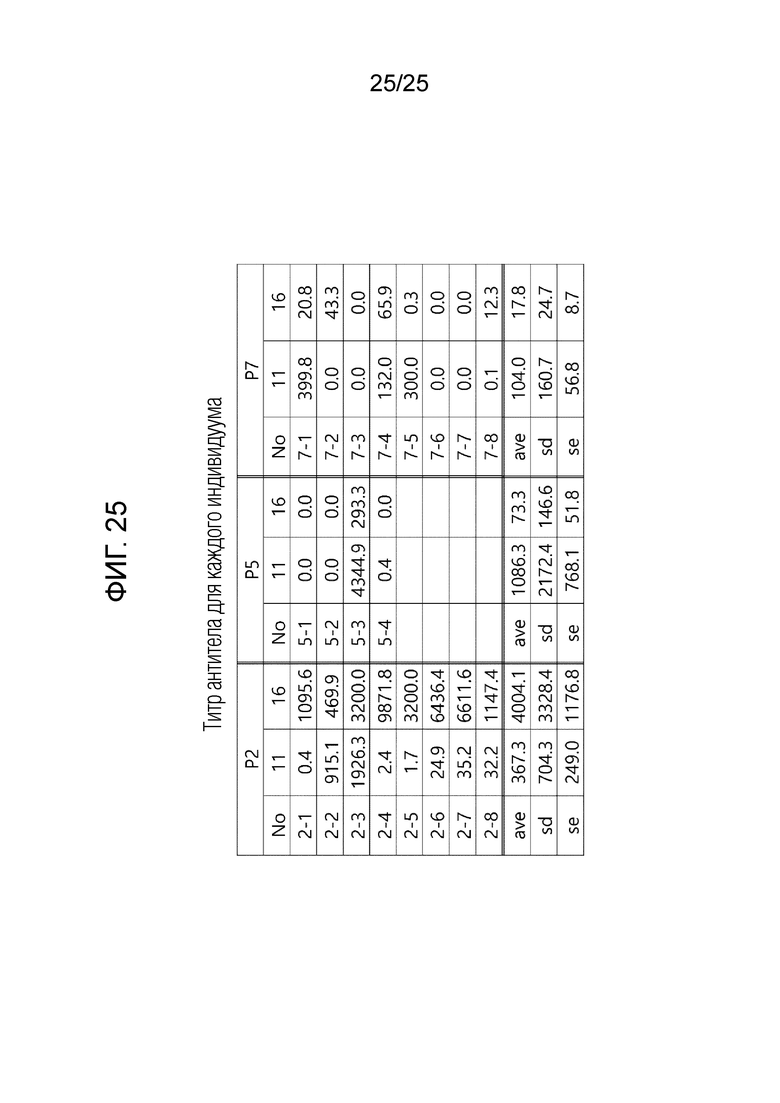
Изобретение относится к области биотехнологии, конкретно к получению модифицированных пептидных эпитопов, и может быть использовано в медицине для лечения ожирения. Предложен пептид, который может распознаваться CD4+ Т-клеткой для индукции гуморального иммунного ответа, при этом пептид состоит из RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAACR, где а обозначает D-форму аланина, (Cha) обозначает L-циклогексилаланин и Z обозначает 6-аминогексановую кислоту. Также предложена композиция для лечения ожирения, содержащая указанный пептид. Изобретение обеспечивает получение пептида, эффективного в снижении массы тела и жировых отложений. 2 н. и 1 з.п. ф-лы, 25 ил., 15 табл., 10 пр.
1. Пептид, который может распознаваться CD4+ Т-клеткой для индукции гуморального иммунного ответа, при этом пептид состоит из RNVPPIFNDVYWIAFZaK(Cha)VAAWTLKAACR (SEQ ID NO: 161),
где а обозначает D-форму аланина, (Cha) обозначает L-циклогексилаланин и Z обозначает 6-аминогексановую кислоту.
2. Композиция для лечения ожирения, содержащая:
терапевтически эффективное количество пептида по п. 1; и
адъюванты.
3. Композиция по п. 2, где адъюванты содержат фосфат алюминия и монофосфорил липид А.
| ИММУНОГЕННЫЙ ГИБРИДНЫЙ ПОЛИПЕПТИД ПРОТИВ ОЖИРЕНИЯ И ВАКЦИННАЯ КОМПОЗИЦИЯ НА ЕГО ОСНОВЕ | 2005 |
|
RU2341534C2 |
| ИММУНОГЕННЫЕ ГИБРИДНЫЕ ПОЛИПЕПТИДЫ ПРОТИВ ОЖИРЕНИЯ И КОМПОЗИЦИЯ ВАКЦИН ПРОТИВ ОЖИРЕНИЯ, СОДЕРЖАЩАЯ ЭТИ ПОЛИПЕПТИДЫ | 2007 |
|
RU2418005C2 |
| ВАКЦИННАЯ КОМПОЗИЦИЯ ПРОТИВ ЗЛОКАЧЕСТВЕННОЙ ОПУХОЛИ НА ОСНОВЕ ПЕПТИДА WT1 ДЛЯ МУКОЗАЛЬНОГО ВВЕДЕНИЯ | 2014 |
|
RU2687026C2 |
| PAKULA A.A | |||
| et al., Genetic analysis of protein stability and function | |||
| Anna | |||
| Rev | |||
| Genet | |||
| Механизм для сообщения поршню рабочего цилиндра возвратно-поступательного движения | 1918 |
|
SU1989A1 |
| TOKURIKI N | |||
| et al., Stability effects of mutations and protein evolvability, Curr | |||
| Opin | |||
| Struct | |||
| Biol., 2009, v.19, n.5, p.596-604; | |||
| HONG S.Y | |||
| et al., | |||
Авторы
Даты
2025-03-11—Публикация
2021-07-21—Подача