Изобретение относится к области биотехнологии и медицины и может быть использовано в качестве основы при разработке лекарственных форм олигонуклеотидных препаратов для терапии аутоиммунных или онкологических заболеваний, а также вирусных инфекций.
Известен способ модификации антисмыслового олигонуклеотида (АСО) для онкомаркер-зависимой деградации целевой мРНК (Drozd V.S., Eldeeb A.A., Kolpashchikov D.M., Nedorezova D.D. Binary Antisense Oligonucleotide Agent for Cancer Marker-Dependent Degradation of Targeted RNA // Nucleic Acid Ther. - 2022. - Vol.32. - № 5. - p. 412-420). Он представляет собой структурную модификацию одноцепочечного аснтисмыслового олигонуклеотида с получением бинарного антисмыслового олигонуклеотида (БиАСО). Способ включает извлечение информации из базы данных о последовательностях биомаркерной РНК и целевой мРНК, определение соответствующей последовательности АСО для целевой мРНК длиной 18-25 нуклеотидов (нт). Далее разделяют АСО на два коротких фрагмента, которые вместе составляют терапевтический модуль БиАСО. В свою очередь каждый фрагмент терапевтического модуля сам по себе сшивают ковалентно через химические линкеры (гексаэтиленгликоль или HEG) с другим АСО, связывающим биомаркеркерную РНК, и также поделенного на два фрагмента, которые вместе составляют сенсорный модуль БиАСО. Таким образом, БиАСО структура состоит из 2 олигонуклеотидов. При гибридизации БиАСО с биомаркерной РНК и целевой РНК образуется структура с четырехсторонним соединением (4-way junction). Ключевым в достижении биомаркер-зависимого расщепления целевой мРНК при использовании БиАСО является подбор термодинамических параметров (температура плавления, энергия Гиббса и др.) так, чтобы короткие фрагменты терапевтического модуля БиАСО могли активироваться и присоединяться к целевой мРНК только будучи связанными с биомаркером. Данный способ предназначен для активации терапевтического действия - деградации мРНК целевого гена с помощью нуклеазы РНКазы H засчет гибридизации БиАСО к целевой мРНК только при наличии РНК биомаркера в инкубационной среде. Этот способ может быть адаптирован и применим в терапии генетических заболеваний, где требуется дифференциация клеток по наличию тех или иных биомаркеров заболевания, или вирусных генетических материалов и др.
Главным недостатком БиАСО структуры является слабая активность расщепления целевой мРНК в связи со снижением аффинности АСО за счет его разделения на два коротких фрагмента. Кроме того, биомаркер-зависимая активация терапевтической функции была продемонстрирована только в условиях близких физиологическим (в растворе) с рекомбинантным прокариотическим ферментом E. Coli. Активированная структура БиАСО представляет собой структуру четырехстороннего соединения олигонуклеотидов, что может ингибировать активность эукариотического фермента РНКазы H внутри клеток. Кроме того, в отличие от предложенного изобретения, в указанном аналоге применялись химически незащищенные олигонуклеотиды, которые не могут быть использованы в работе с клеточными культурами, а также в дальнейшем клиническом применении, в связи с их низкой стабильностью и быстрой деградацией под воздействием внутриклеточных нуклеаз.
Решается задача повышения активности биомаркер-зависимого расщепления целевой мРНК высвобождением одноцепочечного антисмыслового олигонуклеотида (АСО).
Технический результат заключается в обеспечении взаимодействия олигонуклеотидов с биомаркерной РНК внутри клеток.
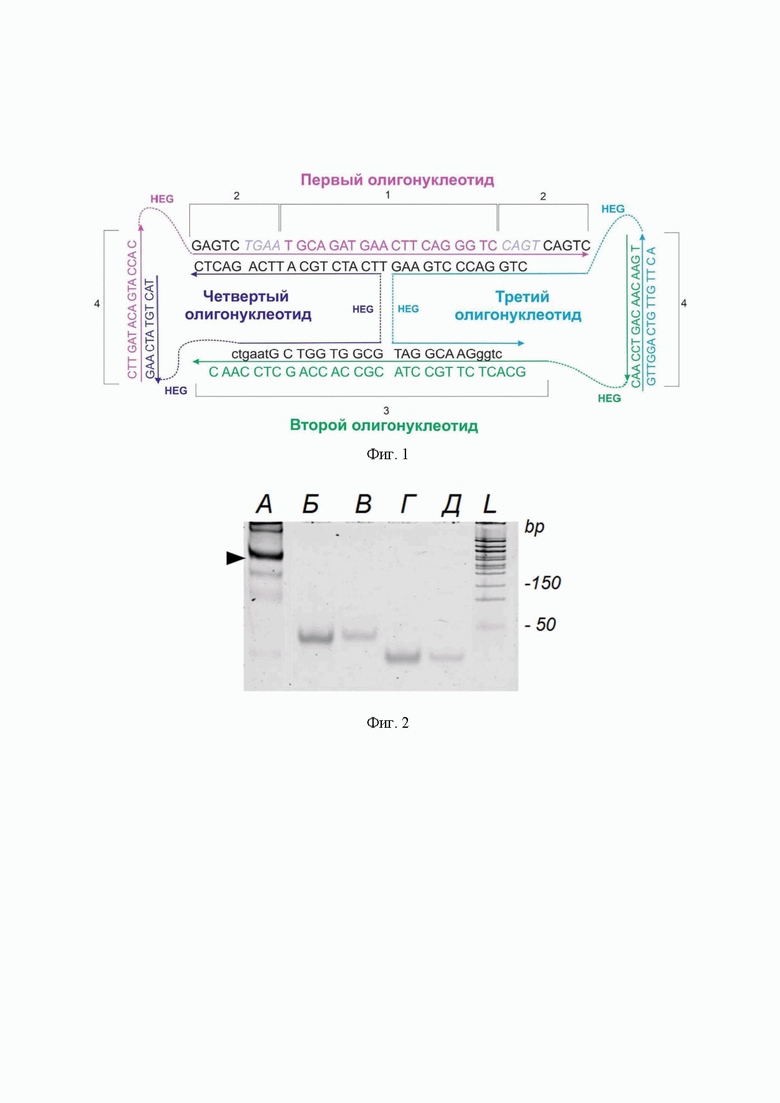
Данный технический результат достигается тем, что способ модификации АСО для онкомаркер-зависимой деградации целевой мРНК, включает извлечение информации из базы данных о последовательностях биомаркерной РНК и целевой мРНК, определение соответствующей последовательности АСО для целевой мРНК длиной 18-25 нуклеотидов (нт), отличается тем, что остов полученного АСО химически модифицируют с помощью тиофосфатов, к АСО добавляют дополнительные 34 нт, из которых, образующие ингибирующие участки и неспособные связываться с целевой мРНК 10 химически модифицированные с помощью 2'-метилирования дезоксирибозы (2'-O-Me) и 8 в виде ДНК нуклеотидов в равных долях располагают симметрично относительно АСО, остальные 16, неспособные связываться ни с одной из нуклеиновых кислот в клетках, связывают с помощью гексаэтиленгликоля (HEG) с полученным АСО с ингибирующими участками, в результате чего получают первый олигонуклеотид, на основе биологически значимого фрагмента биомаркерной РНК строят комплементарную ей последовательность длиной 29 нт, соединяют их с помощью HEG c 16 нт, модифицированными с помощью тиофосфатов, неспособными связываться ни с одной из нуклеиновых кислот в клетках и отличными от первых 16 нт из первого олигонуклеотида, в результате получают второй олигонуклеотид, затем строят комплементарную последовательность к АСО с ингибирующими участками, делят ее на два модифицированных с помощью 2'-O-Me фрагмента равной длины, строят комплементарную последовательность из 16 нт, модифицированных с помощью тиофосфатов, к 16 нт из второго олигонуклеотида, строят комплементарную последовательность к комплементарной последовательности к биомаркерной РНК, разделяют ее на вторые два фрагмента равной длины, модифицированных с помощью 2'-O-Me, последние 16 нт соединяют с первым фрагментом из первой пары, соединяют с первым фрагментом второй пары с помощью двух линкеров HEG, в результате получают третий олигонуклеотид, затем строят комплементарную последовательность к 16 нт из первого олигонуклеотида длиной 12 нт, модифицированных с помощью тиофосфатов, их соединяют со вторым фрагментом первой пары, соединяют со вторым фрагментом второй пары с помощью 2 HEG, в результате получают четвертый олигонуклеотид, после проведения направленного химического синтеза, очистки и лиофилизации полученные 4 олигонуклеотида растворяют в воде предварительно очищенной от РНКаз и ДНКаз, растворы четырех олигонуклеотидов смешивают в равной концентрации в физиологическом буфере (10 мМ Mg2+), полученный раствор нагревают до 95°C, и после охлаждения его до комнатной температуры в течение 4 часов, получают раствор с олигонуклеотидным комплексом, целостность которого проверяют с помощью электрофореза в 10% нативном полиакриламидном геле в течение 90 мин при напряжении на электродах 80 В.
Механизм действия АСО заключается в способности этих молекул связываться с целевой мРНК по комплементарности последовательностей. Связывание приводит к ингибированию процесса трансляции генетической информации в белок. Таким образом, АСО могут блокировать экспрессию конкретного гена, что делает их потенциально полезными в лечении различных заболеваний. Онкомаркер-зависимая активация терапевтической функции АСО позволяет увеличить эффективность терапии онкологий засчет свободного выбора целевого гена-мишени. Этот способ наделяет классическую технологию АСО функцией распознавания биомаркеров нуклеотидной природы и последующей активации терапии только в тех клетках, где есть экспрессия биомаркерных РНК. Способ открывает возможность свободного выбора целевого гена (включая любые жизненно важные гены домашнего хозяйства) для эффективной борьбы с раковыми клетками без значительных побочных эффектов для нормальных клеток, так как терапия активируется только при наличии в клетках мРНК онкогенов или опухолеассоциированных микроРНК.
Второй олигонуклеотид несет в себе две функции: формирует платформу для сборки всех олигонуклеотидов комплекса в общую ДНК структуру и способен связываться со специфическим для конкретного заболевания биомаркером нуклеотидной природы (например, мРНК онкогена KRAS). Высвобождение первого олигонуклеотида из состава комплекса происходит строго после взаимодействия второго олигонуклеотида с биомаркерной РНК в результате их гибридизации по принципу комплементарности. Это дестабилизирует весь комплекс за счет вытеснения второго олигонуклеотида из комплекса.
Первый олигонуклеотид, несущий в себе последовательность терапевтического АСО, высвобождается из олигонуклеотидного комплекса и становится доступным для связывания с мРНК целевого гена и дальнейшего расщепления мРНК РНКазой H только в присутсвии биомаркерной РНК. АСО химически модифицированный с помощью тиофосфатов позволяет сохранять нуклеазную активность фермента РНКазы H и запускать РНКаза H-зависимую деградацию целевой мРНК при гибридизации мРНК с АСО. Ингибирующие участки первого олигонуклеотида олигонуклеотидного комплекса предназначены для исключения несвоевременного выпуска токсичного для клеток терапевтического АСО.
Третий и четвертый олигонуклеотиды химически модифицированы с помощью 2'-O-Me, что позволяет удерживать АСО в составе олигонуклеотидного комплекса. Кроме того, в присутсвии биомаркерной РНК, эти модификации и частичная комплементарность фрагментов первой и второй пары в составе этих олигонуклеотидов позволяют формировать стабильные внутримолекулярные шпилечные структуры, что дестабилизирует удержание первого олигонуклеотида в составе олигонуклеотидного комплекса.
Благодаря интеграции химических модификаций в состав олигонуклеотидного комплекса, способ может быть использован для биомаркер-зависимой деградации целевой мРНК внутри клеток. Химические модификации (тиофосфаты, 2'-O-Me) обеспечивают защиту олигонуклеотидов от деградации с помощью внутриклеточных нуклеаз, тем самым продлевая терапевтическое воздействие олигонуклеотидного комплекса в клетках. Благодаря гибкой адаптации последовательностей олигонуклеотидного комплекса способ имеет потенциальное применение в терапии различных онкологических и генетических заболеваний, а также в персонализированном лечении конкретных пациентов. Способ пригоден для разработки лекарственных форм олигонуклеотидных препаратов в области терапии различных заболеваний, включая онкологию, аутоиммунные заболевания и вирусные инфекции.
Сущность предлагаемого способа поясняется фигурами, где на фиг. 1 представлено схематическое изображение олигонуклеотидного комплекса, на фиг. 2 - результат электрофореза в нативном полиакриламидном геле, на фиг. 3 - результаты подавления флуоресценции клеток с помощью олигонуклеотидного комплекса, а на фиг. - 4 результаты количественного ПЦР относительной экспрессии целевой мРНК.
После извлечения информации из базы данных о последовательностях биомаркерной РНК (например, мРНК онкогена, микроРНК или др.) и целевой мРНК, используя известные базы данных (например, NCBI) в зависимости от исследовательской или биомедицинской задачи. Далее определяют соответствующую последовательность АСО для целевой мРНК длиной 18-25 нуклеотидов (нт), используя предсказательные программы, которые учитывают вторичные структуры мРНК, GC-состав, длину АСО и др. (например, программа PFREED). Из предложенных программой вариантов, выбирают 1 вариант АСО для последующей работы. Опционально проверяют неспецифические взаимодействия, разработанного АСО, с нецелевыми генами с помощью предсказательных программ (например, BLAST).
Остов полученного АСО химически модифицируют с помощью тиофосфатов, к АСО добавляют дополнительные 34 нт, из которых, образующие ингибирующие участки и неспособные связываться с целевой мРНК 10 химически модифицированные с помощью 2'-метилирования дезоксирибозы (2'-O-Me) и 8 в виде ДНК нуклеотидов в равных долях располагают симметрично относительно АСО, остальные 16, неспособные связываться ни с одной из нуклеиновых кислот в клетках, связывают с помощью гексаэтиленгликоля (HEG) с полученным АСО с ингибирующими участками, в результате чего получают первый олигонуклеотид.
Далее на основе биологически значимого фрагмента биомаркерной РНК строят комплементарную ей последовательность длиной 29 нт, соединяют их с помощью HEG c 16 нт, модифицированными с помощью тиофосфатов, неспособными связываться ни с одной из нуклеиновых кислот в клетках и отличными от первых 16 нт из первого олигонуклеотида, в результате получают второй олигонуклеотид.
Затем строят комплементарную последовательность к АСО с ингибирующими участками, делят ее на два модифицированных с помощью 2'-O-Me фрагмента равной длины, строят комплементарную последовательность из 16 нт, модифицированных с помощью тиофосфатов, к 16 нт из второго олигонуклеотида, строят комплементарную последовательность к комплементарной последовательности к биомаркерной РНК, разделяют ее на вторые два фрагмента равной длины, модифицированных с помощью 2'-O-Me, последние 16 нт соединяют с первым фрагментом из первой пары, соединяют с первым фрагментом второй пары с помощью 2 HEG, в результате получают третий олигонуклеотид.
Затем строят комплементарную последовательность к 16 нт из первого олигонуклеотида длиной 12 нт, модифицированных с помощью тиофосфатов, их соединяют со вторым фрагментом первой пары, соединяют со вторым фрагментом второй пары с помощью 2 HEG, в результате получают четвертый олигонуклеотид.
После проведения направленного химического синтеза, очистки и лиофилизации, полученные 4 олигонуклеотида растворяют в воде предварительно очищенной от РНКаз и ДНКаз. Растворы четырех олигонуклеотидов смешивают в равной концентрации в физиологическом буфере (при содержании 10 мМ Mg2+), полученный раствор нагревают до 95°C, и после охлаждения его до комнатной температуры в течение 4 часов, получают раствор с олигонуклеотидным комплексом, целостность которого проверяют с помощью электрофореза в 10% нативном полиакриламидном геле в течение 90 мин при напряжении на электродах 80 В.
Пример. В качестве целевой мРНК был выбран ген зеленого флуоресцентного белка (GFP). Для терапии рака модель мРНК GFP должна быть заменена на мРНК жизненно важного гена домашнего хозяйства. Цель инактивации таких генов с помощью АСО - вызвать апоптоз раковых клеток зависимым от биомаркеров способом. В качестве биомаркерной РНК был выбран фрагмент длиной 29 нт мРНК гена KRAS, который является протоонкогеном из семейства RAS, сверхэкспрессируется при некоторых типах опухолей, включая аденокарциному легкого, муцинозную аденому, протоковую карциному поджелудочной железы и колоректальный рак. Далее были разработаны 4 олигонуклеотида, как указано в описываемом способе. Все последовательности олигонуклеотидов, использованные в примере, представлены в таблице 1.
Таблица 1. Список разработанных олигонуклеотидов

N обычный - тиофосфаты
N подчеркнутый - 2'-O-Me N курсив - ДНК нуклеотиды незащищенные /HEG/ - гексаэтиленгликолевый линкер В данном примере синтезированный в соответствии с предлагаемым способом олигонуклеотидный комплекс, состоит (фиг. 1) из последовательности исходного АСО для целевой мРНК 1, ингибирующих участков 2, последовательности комплиментарной к биомаркерной РНК 3, последовательностей, не способных связываться ни с одной из нуклеиновых кислот в клетках 4. Сборка комплекса проводилась путем смешивания 4 олигонуклеотидов в эквимолярной концентрации (1 мкМ) в буфере (KCl 150 мМ, HEPES 50 мМ и pH 7.4, NaCl 15 мМ, MgCl2 10 мМ). Смесь в пробирке помещалась в предварительно разогретую до 95°C тару с 500 мл воды и оставалась остывать в течение 4 часов до комнатной температуры. Результат сборки оценивались с помощью метода электрофореза в 10% нативном полиакриламидном геле (ПААГ) (AABA 40% 2,5 мл, TBE 10X 1 мл, TEMED 10 мкл, APS 10% 100 мкл, MgCl2 50 нМ) (80 В, 120 мин). Состав буфера для геля TBE 10X: 890 мМ Трис-(гидроксиметил) аминометан, 890 мМ борная кислота, 20 мМ ЭДТА, pH 8.3. Затем гель был окрашен в течение 5 мин с этидиумом бромидом. Результаты электрофореза в нативном ПААГ, представлены на фиг. 2 где А - черной стрелкой показан олигонуклеотидный комплекс, Б - первый олигонуклеотид, В - второй олигонуклеотид, Г - третий олигонуклеотид, Д - четвертый олигонуклеотид, L - ДНК маркер 50 bp+.
Далее проводилась оценка биомаркер-зависимой функции олигонуклеотидного комплекса в клетках миелоидного лейкоза K562 с искусственно индуцированным геном зеленого флуоресцентного белка (GFP). Оценка проводилась путем тестирования подавления экспрессии целевого гена GFP с помощью ОТ-кПЦР (600 тыс. клеток на лунку) и проточной цитофлуориметрии (50 тыс. клеток на лунку) в результате 24-часовой трансфекции олигонуклеотидного комплекса (100 нМ) и контрольных образцов в клеточные линии. Трансфекция производилась с помощью липосомальных наночастиц (GeneJector, по протоколу производителя Молекта).
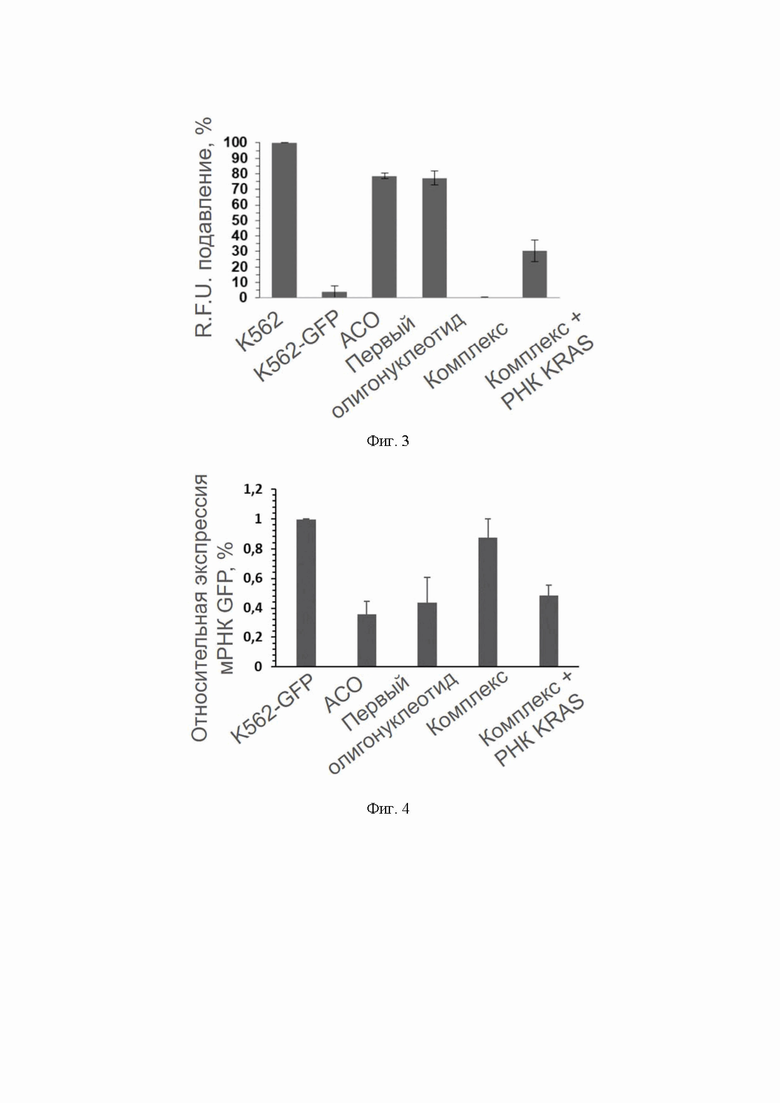
Относительные единицы флуоресценции (R.F.U.) оценивались путем определения медианы интенсивности сигнала внутренней флуоресценции (FITC) в популяциях клеток K562 (учитывались только синглеты с нормальной морфологией), с последующей Min-Max нормализацией (на графике представлено три независимых эксперимента со стандартным отклонением). По результатам проточной цитофлуорометрии подавление общей флуоресценции клеток с помощью олигонуклеотидного комплекса составило 0,5%, в то время как в присутствии биомаркера РНК KRAS подавление составило 30%, что доказывает биомаркер-зависимую активацию подавления активности целевого гена GFP (фиг. 3).
В результате ОТ-кПЦР анализа после трансфекции олигонуклеотидного комплекса экспрессия мРНК GFP составляла 87% (подавление - 23%), в то время как с биомаркером РНК KRAS экспрессия мРНК GFP составила 49% (подавление 51%) (фиг. 4). Каждый столбец графика соответствует уровню экспрессии мРНК GFP по отношению к нетрансфецируемому контролю - K562-GFP. Уровень экспрессии мРНК GFP был нормализован на ген домашнего хозяйства HPRT. Результат демонстрирует двукратное снижение экспрессии целевой мРНК гена GFP в клеточных условиях с помощью комплекса в присутствии фрагмента РНК KRAS по сравнению с образцом в его отсутствии. Таким образом, данные, полученные для комплекса в клеточной линии миелоидного лейкоза K562 свидетельствуют о биомаркер-зависимом распаде комплекса и высвобождении АСО.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Способ модификации антисмыслового олигонуклеотида для
онкомаркер-зависимого деградации целевой мРНК.xml" softwareName="WIPO
Sequence" softwareVersion="2.3.0" productionDate="2025-02-02">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2024117050 </ApplicationNumberText>
<FilingDate>2024-06-20</FilingDate>
</ApplicationIdentification>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2024117050 </ApplicationNumberText>
<FilingDate>2024-06-20</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ITMO University</ApplicantName>
<InventionTitle languageCode="ru">Способ модификации антисмыслового
олигонуклеотида для онкомаркер-зависимой деградации целевой
мРНК</InventionTitle>
<SequenceTotalQuantity>10</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Phosphorothioate</INSDQualifier_value>
<NonEnglishQualifier_value>Тиофосфат</NonEnglishQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcagatgaacttcagggtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>54</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..54</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_binding</INSDFeature_key>
<INSDFeature_location>15^16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q28">
<INSDQualifier_name>bound_moiety</INSDQualifier_name>
<INSDQualifier_value>Hexaethylenglycol
(HEG)</INSDQualifier_value>
<NonEnglishQualifier_value>Гексаэтиленгликоль</NonEnglishQualif
ier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location><20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Phosphorotioate</INSDQualifier_value>
<NonEnglishQualifier_value>Тиофосфат</NonEnglishQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>25..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Phosphorotioate</INSDQualifier_value>
<NonEnglishQualifier_value>Тиофосфат</NonEnglishQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>49..54</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Phosphorotioate</INSDQualifier_value>
<NonEnglishQualifier_value>Тиофосфат</NonEnglishQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttgatacagtaccacgagtctgaatgcagatgaacttcagggtccagt
cagtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Phosphorothioate</INSDQualifier_value>
<NonEnglishQualifier_value>Тиофосфат</NonEnglishQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_binding</INSDFeature_key>
<INSDFeature_location>16^17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q34">
<INSDQualifier_name>bound_moiety</INSDQualifier_name>
<INSDQualifier_value>Hexaethylenglycol
(HEG)</INSDQualifier_value>
<NonEnglishQualifier_value>Гексаэтиленгликоль</NonEnglishQualif
ier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgaacaacagtccaacgcactcttgcctacgccaccagctccaac</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_binding</INSDFeature_key>
<INSDFeature_location>16^17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q36">
<INSDQualifier_name>bound_moiety</INSDQualifier_name>
<INSDQualifier_value>Hexaethyleneglycoll
(HEG)</INSDQualifier_value>
<NonEnglishQualifier_value>Гексаэтиленгликоль</NonEnglishQualif
ier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_binding</INSDFeature_key>
<INSDFeature_location>29^30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q37">
<INSDQualifier_name>bound_moiety</INSDQualifier_name>
<INSDQualifier_value>Hexaethyleneglycoll
(HEG)</INSDQualifier_value>
<NonEnglishQualifier_value>Гексаэтиленгликоль</NonEnglishQualif
ier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location><16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Phosphorothioate</INSDQualifier_value>
<NonEnglishQualifier_value>Тиофосфат</NonEnglishQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>>17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2'-O-Methylation</INSDQualifier_value
>
<NonEnglishQualifier_value>2'-О-Метил</NonEnglishQualifier
_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttggactgttgttcactggaccctgaagtggcaagggtc</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location><12</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q41">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Phosphorothioate</INSDQualifier_value>
<NonEnglishQualifier_value>Тиофосфат</NonEnglishQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>>13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2'-O-Methylation</INSDQualifier_value
>
<NonEnglishQualifier_value>2'-O-Метилирование</NonEnglishQ
ualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_binding</INSDFeature_key>
<INSDFeature_location>12^13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q43">
<INSDQualifier_name>bound_moiety</INSDQualifier_name>
<INSDQualifier_value>Hexaethylenglycol
(HEG)</INSDQualifier_value>
<NonEnglishQualifier_value>Гексаэтиленгликоль</NonEnglishQualif
ier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_binding</INSDFeature_key>
<INSDFeature_location>28^29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q44">
<INSDQualifier_name>bound_moiety</INSDQualifier_name>
<INSDQualifier_value>Hexaethylenglycol
(HEG)</INSDQualifier_value>
<NonEnglishQualifier_value>Гексаэтилентгликоль</NonEnglishQuali
fier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tactgtatcaagctgaatgctggtggcgttcatctgcattcagactc</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q45">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttatggcgacccgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accctttccaaatcctcagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q47">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgtaaacggccacaagttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagtcgtgctgcttcatgtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q51">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>2'-O-Methylation</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttggagctggtggcgtaggcaagagtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления вируса Nipah методом, основанным на применении дезоксирибозима 10-23 | 2023 |
|
RU2816271C1 |
| Способ получения мышиной модели для изучения миодистрофии Дюшенна и вариантов ее терапии | 2023 |
|
RU2815936C1 |
| Олигонуклеотиды для диагностики эпилепсии методом количественной ПЦР | 2023 |
|
RU2815113C1 |
| КОМБИНИРОВАННАЯ ТЕРАПИЯ ГЕПАТОЦЕЛЛЮЛЯРНОЙ КАРЦИНОМЫ НА ОСНОВЕ СОРАФЕНИБА | 2022 |
|
RU2800071C1 |
| АНТИСМЫСЛОВЫЕ ОЛИГОНУКЛЕОТИДЫ, НАЦЕЛЕННЫЕ НА ГЕН CAV3.1, И ИХ ПРИМЕНЕНИЯ | 2022 |
|
RU2839721C2 |
| СПОСОБ АМПЛИФИКАЦИИ И ИДЕНТИФИКАЦИИ НУКЛЕИНОВЫХ КИСЛОТ | 2019 |
|
RU2811465C2 |
| Способ прогностической оценки развития гепатоцеллюлярной карциномы на основе определения полиморфизма гена человека IFNAR-1 и набор олигодезоксирибонуклеотидных праймеров | 2022 |
|
RU2811763C1 |
| Набор реагентов для определения малых рибонуклеиновых кислот (микроРНК) при диагностике множественной миеломы | 2024 |
|
RU2836054C1 |
| Способ определения делеции 7 экзона в гене SMN1 с определением количества копий гена SMN2 в образцах ДНК с помощью цифровой полимеразной цепной реакции | 2024 |
|
RU2838673C1 |
| Способ выявления рекомбинантных форм вируса гепатита C и мутаций лекарственной устойчивости вируса гепатита С к препаратам прямого противовирусного действия методом полимеразной цепной реакции с последующим секвенированием | 2023 |
|
RU2824565C1 |
Настоящее изобретение относится к области биотехнологии. Описан способ модификации антисмыслового олигонуклеотида (АСО) для онкомаркер-зависимой деградации целевой мРНК путём смешивания четырёх описанных олигонуклеотидов с получением олигонуклеотидного комплекса. Технический результат заключается в обеспечении взаимодействия олигонуклеотидов с биомаркерной РНК внутри клеток. 4 ил., 1 табл., 1 пр.
Способ модификации антисмыслового олигонуклеотида (АСО) для онкомаркер-зависимой деградации целевой мРНК, включающий извлечение информации из базы данных о последовательностях биомаркерной РНК и целевой мРНК, определение соответствующей последовательности АСО для целевой мРНК длиной 18-25 нуклеотидов (нт), отличающийся тем, что остов полученного АСО химически модифицируют с помощью тиофосфатов, к АСО добавляют дополнительные 34 нт, из которых образующие ингибирующие участки и неспособные связываться с целевой мРНК 10 химически модифицированные с помощью 2'-метилирования дезоксирибозы (2'-O-Me) и 8 в виде ДНК нуклеотидов в равных долях располагают симметрично относительно АСО, остальные 16, неспособные связываться ни с одной из нуклеиновых кислот в клетках, связывают с помощью гексаэтиленгликоля (HEG) с полученным АСО с ингибирующими участками, в результате чего получают первый олигонуклеотид, на основе биологически значимого фрагмента биомаркерной РНК строят комплементарную ей последовательность длиной 29 нт, соединяют их с помощью HEG c 16 нт, модифицированными с помощью тиофосфатов, неспособными связываться ни с одной из нуклеиновых кислот в клетках и отличными от первых 16 нт из первого олигонуклеотида, в результате получают второй олигонуклеотид, затем строят комплементарную последовательность к АСО с ингибирующими участками, делят ее на два модифицированных с помощью 2'-O-Me фрагмента равной длины, строят комплементарную последовательность из 16 нт, модифицированных с помощью тиофосфатов, к 16 нт из второго олигонуклеотида, строят комплементарную последовательность к комплементарной последовательности к биомаркерной РНК, разделяют ее на вторые два фрагмента равной длины, модифицированные с помощью 2'-O-Me, последние 16 нт соединяют с первым фрагментом из первой пары, соединяют с первым фрагментом второй пары с помощью 2 HEG, в результате получают третий олигонуклеотид, затем строят комплементарную последовательность к 16 нт из первого олигонуклеотида длиной 12 нт, модифицированных с помощью тиофосфатов, их соединяют со вторым фрагментом первой пары, соединяют со вторым фрагментом второй пары с помощью 2 HEG, в результате получают четвертый олигонуклеотид, после проведения направленного химического синтеза, очистки и лиофилизации полученные 4 олигонуклеотида растворяют в воде, предварительно очищенной от РНКаз и ДНКаз, растворы четырех олигонуклеотидов смешивают в равной концентрации в физиологическом буфере с содержанием хлорида магния в концентрации 10 мМ, полученный раствор нагревают до 95°C и после охлаждения его до комнатной температуры в течение 4 часов получают раствор с олигонуклеотидным комплексом, целостность которого проверяют с помощью электрофореза в 10% нативном полиакриламидном геле в течение 90 мин при напряжении на электродах 80 В.
| Drozd V.S | |||
| et al | |||
| Binary Antisense Oligonucleotide Agent for Cancer Marker-Dependent Degradation of Targeted RNA | |||
| Nucleic Acid Ther,2022, v.32 (5), p | |||
| Способ применения поваренной соли в нагревательной закалочной ванне при высоких температурах | 1923 |
|
SU412A1 |
| US 20080255066 A1, 16.10.2008 | |||
| КОМПОЗИЦИЯ ДЛЯ ЛЕЧЕНИЯ РАКА, АССОЦИИРОВАННОГО С ИНФЕКЦИЕЙ HPV | 2017 |
|
RU2664466C1 |
Авторы
Даты
2025-04-29—Публикация
2024-06-20—Подача