Область техники
Изобретение относится к областям молекулярных биотехнологии и медицины и касается мышиной модели миодистрофии Дюшенна, несущей мутацию в 51-м экзоне гена дистрофина мыши Dmd. Изобретение может быть использовано для детального изучения патогенеза и разработки терапии миодистрофии Дюшенна.
Изобретение создано при финансовой поддержке Министерства Науки и Высшего образования Российской Федерации в рамках Соглашения № 075-15-2019-1661 от 31.10.2019 на базе Центра высокоточного редактирования и генетических технологий для биомедицины ИБГ РАН (ЦВРГТБ ИБГ РАН).
Уровень техники
Миодистрофия Дюшенна - моногенное X-сцепленное заболевание, вызываемое мутациями в гене дистрофина DMD. Носители мутации мужского пола, обладающие единственной Х-хромосомой, становятся инвалидами в начале второго десятилетия жизни, а средняя продолжительность их жизни составляет около 25 лет, хотя к этому моменту больные уже не дышат самостоятельно. Болезнь довольно распространенная - мутацию в гене дистрофина DMD имеет один новорожденный мальчик из 5000. Мутации, вызывающие миодистрофию Дюшенна, неравномерно распределены по последовательности гена: наиболее часто мутации затрагивают первые экзоны, а также экзоны с 43 по 55-й (Chen C, Ma H, Zhang F, Chen L, Xing X, Wang S, Zhang X, Luo Y. Screening of Duchenne muscular dystrophy (DMD) mutations and investigating its mutational mechanism in Chinese patients. PLoS One. 2014 Sep 22;9(9):e108038. doi: 10.1371/journal.pone.0108038). Несмотря на то, что миодистрофия Дюшенна - моногенное заболевание, в разработке генной терапии против неё есть определенные сложности. Главная сложность связана с тем, что кодирующая последовательность дистрофина слишком велика (около 14 тысяч пар оснований), чтобы быть эффективно доставленной в клетки. В связи с этим ведется разработка генно-терапевтических подходов, направленных на альтернативный сплайсинг гена дистрофина DMD с пропуском экзонов, содержащих мутации, и восстановление нарушенной рамки считывания. Для испытания такой терапии требуются адекватные мышиные модели, в которых мутации имитировали бы распространенные мутации человека.
Самая распространенная линия мышей, используемая как модель миодистрофии Дюшенна - линия mdx. У данной линии мутация, приводящая к сдвигу рамки считывания и образованию преждевременного стоп-кодона, расположена в 23-м экзоне. Эта модель характеризуется крайне мягкими проявлениями, заметными только на гистологическом уровне. У мышей, у которых полностью отсутствует локус гена мыши Dmd (Kudoh H, Ikeda H, Kakitani M, Ueda A, Hayasaka M, Tomizuka K, Hanaoka K. A new model mouse for Duchenne muscular dystrophy produced by 2.4 Mb deletion of dystrophin gene using Cre-loxP recombination system. Biochem Biophys Res Commun. 2005 Mar 11;328(2):507-16. doi: 10.1016/j.bbrc.2004.12.191) исключена вероятность смягчения состояния из-за механизма пропуска экзонов и наблюдается наиболее тяжелый фенотип, которого теоретически можно добиться, внося мутации в ген дистрофина мыши Dmd. Однако для тестирования генной терапии, манипулирующей сплайсингом, они также не являются подходящей моделью. Для большей выраженности фенотипа были получены мыши с мутациями одновременно в дистрофине и его гомологе - утрофине. Патологические процессы у таких мышей становятся заметны в возрасте 4-х месяцев. Существуют и другие линии мышей, у которых патологическая картина близкая к картине у людей с миодистрофией Дюшенна достигается за счёт нокаута второго гена (Yucel N, Chang AC, Day JW, Rosenthal N, Blau HM. Humanizing the mdx mouse model of DMD: the long and the short of it. NPJ Regen Med. 2018 Feb 16;3:4. doi: 10.1038/s41536-018-0045-4. Erratum in: NPJ Regen Med. 2020 Dec 21;5(1):25). Такие модели, однако, ограниченно годятся для оценки эффективности генной терапии, направленной на восстановление функций гена дистрофина DMD.
Краткое описание изобретения
Целью заявляемого изобретения было получение такой модели мыши, которая сочетала бы в себе выраженный фенотип и пригодность для испытаний современных подходов генной терапии - т.е. небольшую мутацию примерно в области 50-го экзона, поскольку это место с наибольшей вероятностью мутаций у пациентов.
Для внесения мутации, приводящей к сдвигу рамки считывания и образованию преждевременного стоп-кодона, в геном мыши была использована система CRISPR/Cas9, состоящая из белка spCas9 и гидовой РНК (сгРНК), PAM-сайт которой расположен в 51-м экзоне гена дистрофина мыши Dmd.
Задачей заявляемого изобретения является создание мышиной модели миодистрофии Дюшенна путем внесения мутаций в 51-й экзон гена дистрофина мыши Dmd. На этой модели можно тестировать прототипы генной терапии, направленные на альтернативный сплайсинг в этом районе.
Задача решается тем, что с использованием системы CRISPR/Cas9 получены две новых линии генетически модифицированных мышей, содержащих в геноме мутацию (инсерцию - вставку одного нуклеотида) в гене дистрофина мыши Dmd, приводящие к образованию укороченной формы белка дистрофина, для чего:
1) Была получена плазмидная конструкция pSK-T7+sgDmd для in vitro синтеза гидовой РНК (называемой также одиночной гидовой РНК, сгРНК, sgRNA), несущая вектор pSK-T7 последовательностью SEQ ID NO: 1 и кодирующая сгРНК (гидовую РНК) последовательностью SEQ ID NO: 2 против соответствующего участка гена дистрофина мыши Dmd, под контролем T7 промотора.
2) Был разработан способ получения конструкции pSK-T7+sgDmd, характеризующийся следующими стадиями:
a) биоинформатический анализ последовательности гена дистрофина мыши Dmd и подбор сгРНК (гидовой РНК) в области экзона 51;
b) клонирование соответствующей последовательности сгРНК (гидовой РНК) в вектор pSK-T7 последовательностью SEQ ID NO: 1.
3) Способ получения линий мышей с мутациями в 51-м экзоне гена дистрофина мыши Dmd, характеризующийся следующими стадиями:
a) c помощью конструкции pSK-T7+sgDmd по п.1 осуществляют синтез in vitro сгРНК (гидовой РНК) последовательностью SEQ ID NO: 2 против 51-го экзона гена дистрофина мыши Dmd, а также мРНК гена spCas9;
b) далее проводят микроинъекцию смеси сгРНК и мРНК spCas9 из пункта (a) в цитоплазму оплодотворенной яйцеклетки мыши гибрида первого поколения F1(CBA×C57BL/6);
c) выявляют жизнеспособных зигот;
d) пересаживают выживших зигот из пункта (c) псевдобеременным самкам-реципиентам, имеющим копулятивную пробку после ссаживания с вазэктомированными самцами;
e) получают новорожденных мышей на 19 день после пересадки зигот из пункта (d);
f) проводят анализ наличия мутации через 8–14 дней после рождения мышей (e), путем амплификации целевого участка гена дистрофина мыши Dmd с двумя праймерами: Dmd–check-f олигонуклеотидом для генотипирования, имеющего последовательность 5'-TGTTCTCTGGTGTACTGCCT-3' (SEQ ID NO: 3) и Dmd-check-r олигонуклеотидом для генотипирования, имеющего последовательность 5'-TGTTCACAGAGAAAAGGTAGCC-3' (SEQ ID NO: 4) – с последующим секвенированием фрагмента;
g) отбирают мышей, несущих целевую мутацию;
h) проводят скрещивание мышей из пункта (g) с мышами дикого типа для получения самок-гетерозигот или мутантных самцов, несущих целевую мутацию.
Технический результат изобретения: разработан способ получения линии трансгенных мышей с мутациями в 51-м экзоне гена дистрофина мыши Dmd, приводящими к сдвигу рамки считывания, и выведены 2 соответствующие линии мышей с отличающимся фенотипом.
Изобретение иллюстрируется десятью фигурами и перечнем использованных последовательностей.
Краткое описание чертежей
Фигура 1. Электрофорез ПЦР-фрагмента для in vitro синтеза сгРНК. Видно, что в ходе ПЦР амплифицировался единственный продукт, его длина соответствует ожидаемой (149 п.н.).
Фигура 2. Электрофорез сгРНК, синтезированной in vitro. Видно, что в ходе реакции синтезировался единственный продукт, его длина соответствует ожидаемой (120 п.н.).
Фигура 3. Электрофорез мРНК Cas9. Видно, что в ходе реакции синтезировался единственный продукт.
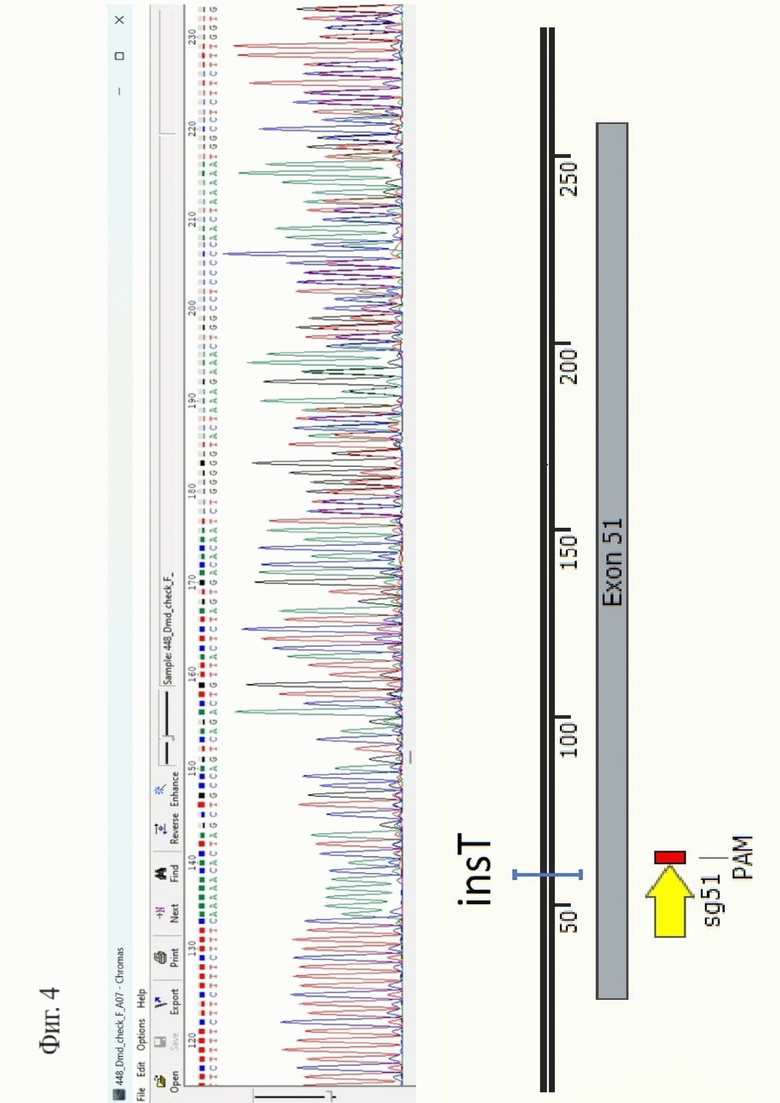
Фигура 4. Хроматограмма сиквенса и схематическое изображение события, вызвавшего сдвиг рамки считывания у основателя линии N1.
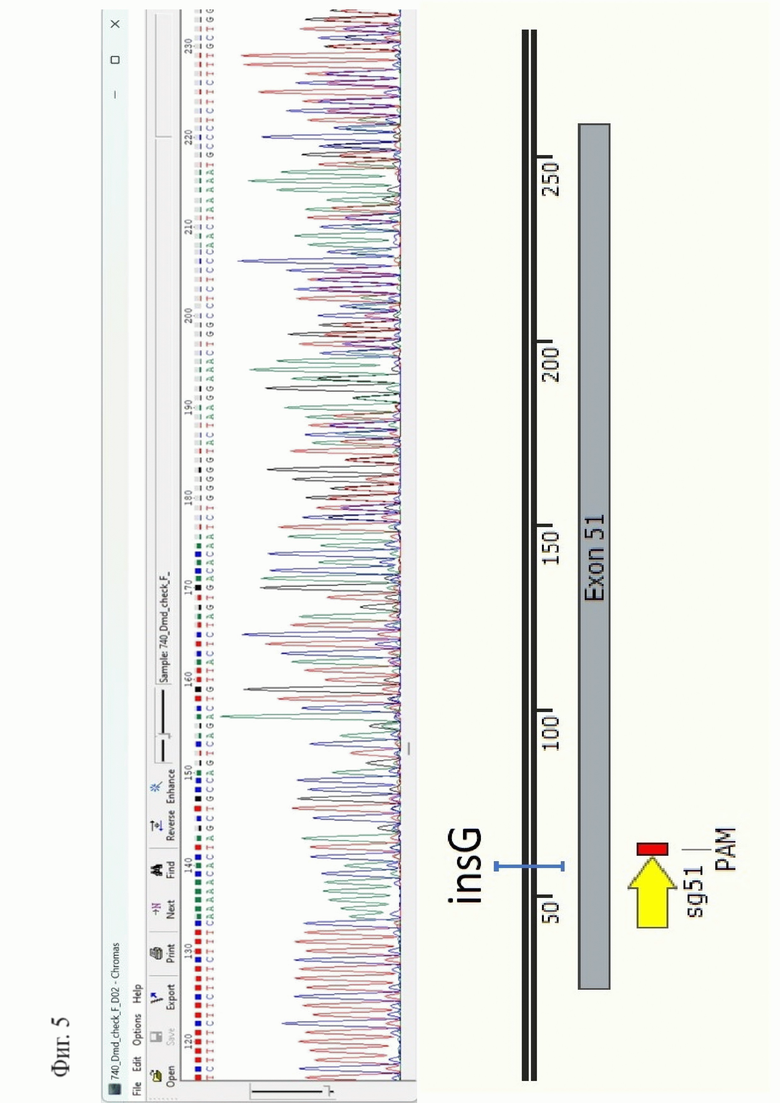
Фигура 5. Хроматограмма сиквенса и схематическое изображение события, вызвавшего сдвиг рамки считывания у основателя линии N2.
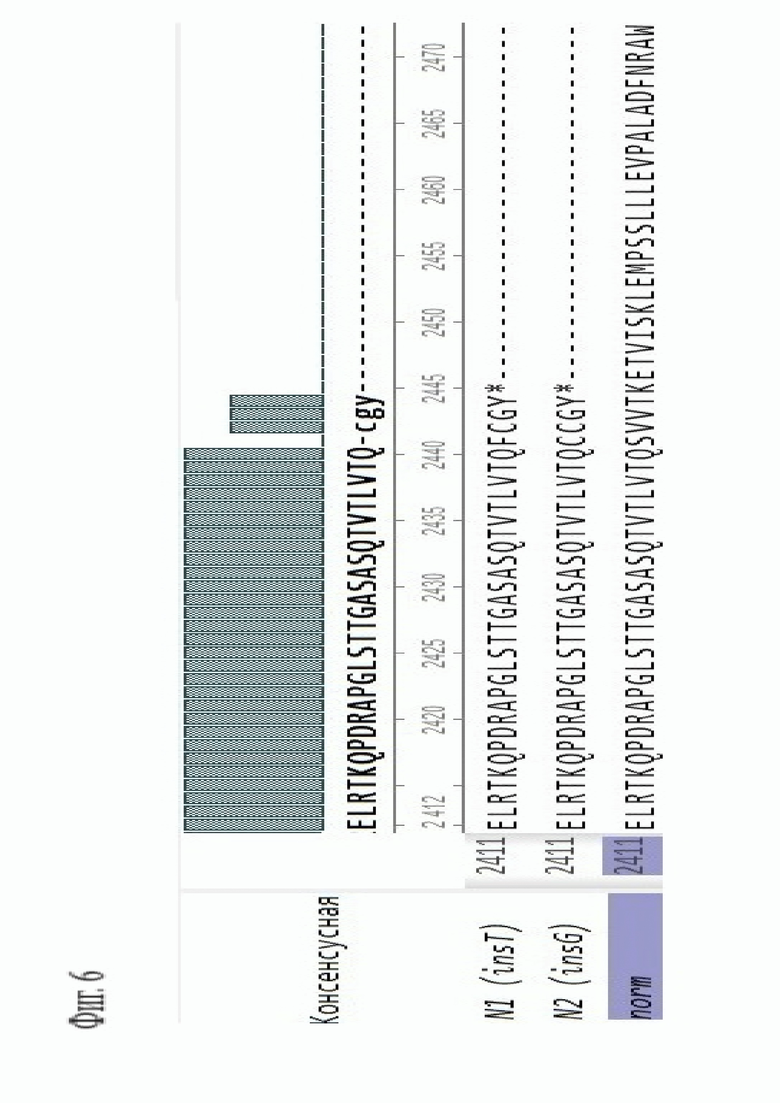
Фигура 6. Сравнение белковых последовательностей нормального (norm) белка дистрофина и мутантных вариантов линий N1 (вариант insT) и N2 (вариант insG).
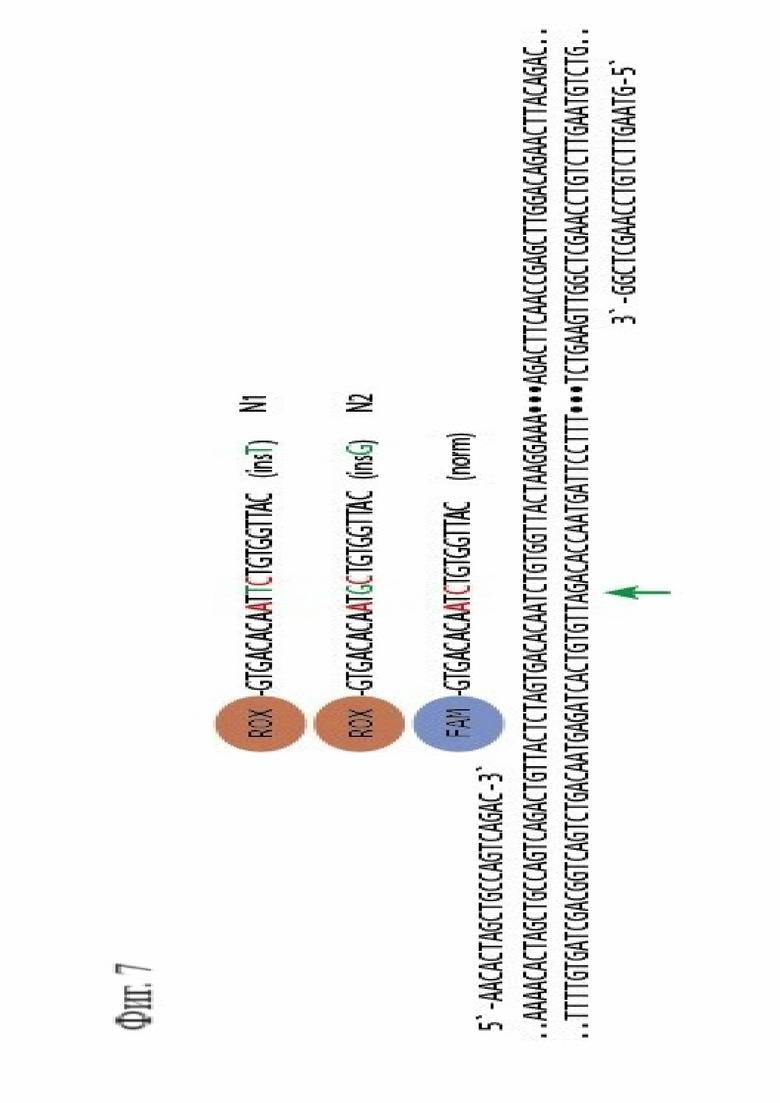
Фигура 7. Система для генотипирования представителей линий N1 (вариант insT) и N2 (вариант insG). Красным цветом отмечены замкнутые нуклеотиды (LNA). Зелёным цветом отмечены инсерции, определяемые зондами, также стрелкой обозначено место инсерции.
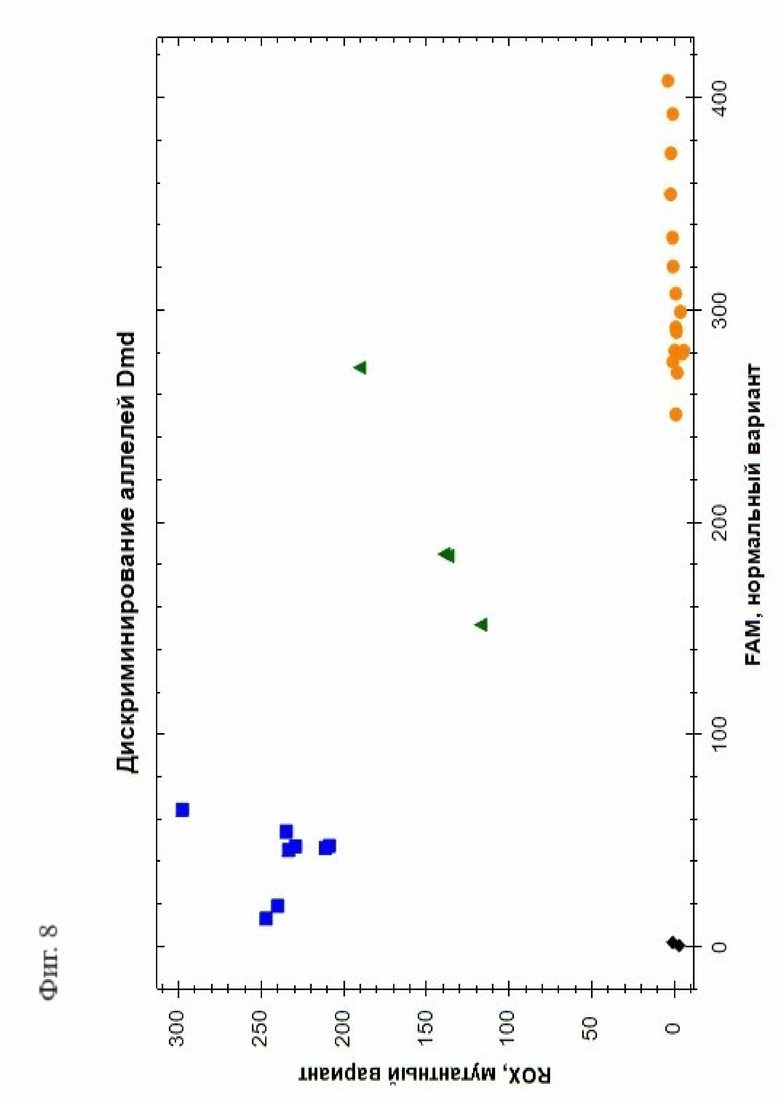
Фигура 8. Пример генотипирования последующих поколений методом ПЦР в реальном времени. Определение аллелей потомства линии N1. Синими точками обозначены пробы, содержащие только мутантные аллели, оранжевым - только дикие, зелёным - гетерозиготы.
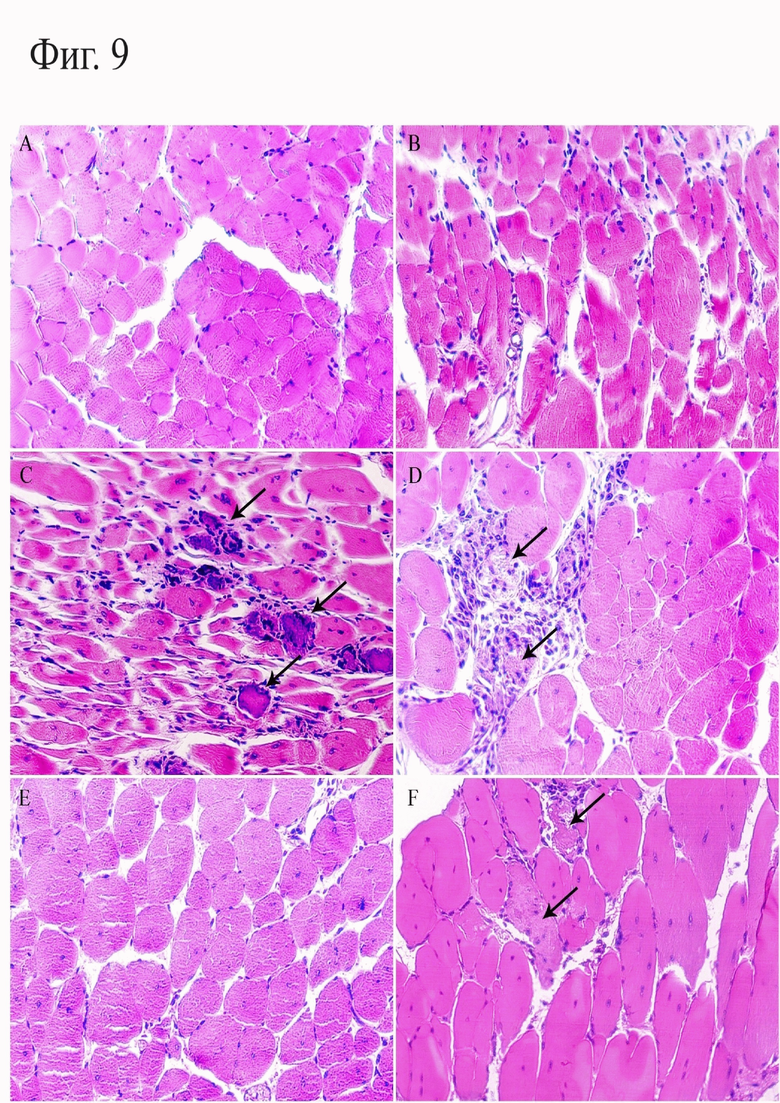
Фигура 9. Поперечные срезы мышц, окраска гематоксилином и эозином, 10х. А - икроножные мышцы самца дикого типа, нормальное строение. В - межреберные мышцы самца дикого типа, нормальное строение. С - икроножные мышцы самца линии N2. D - межреберные мышцы мыши линии N2. E - икроножные мышцы самца линии N1. F - межреберные мышцы самца линии N1.
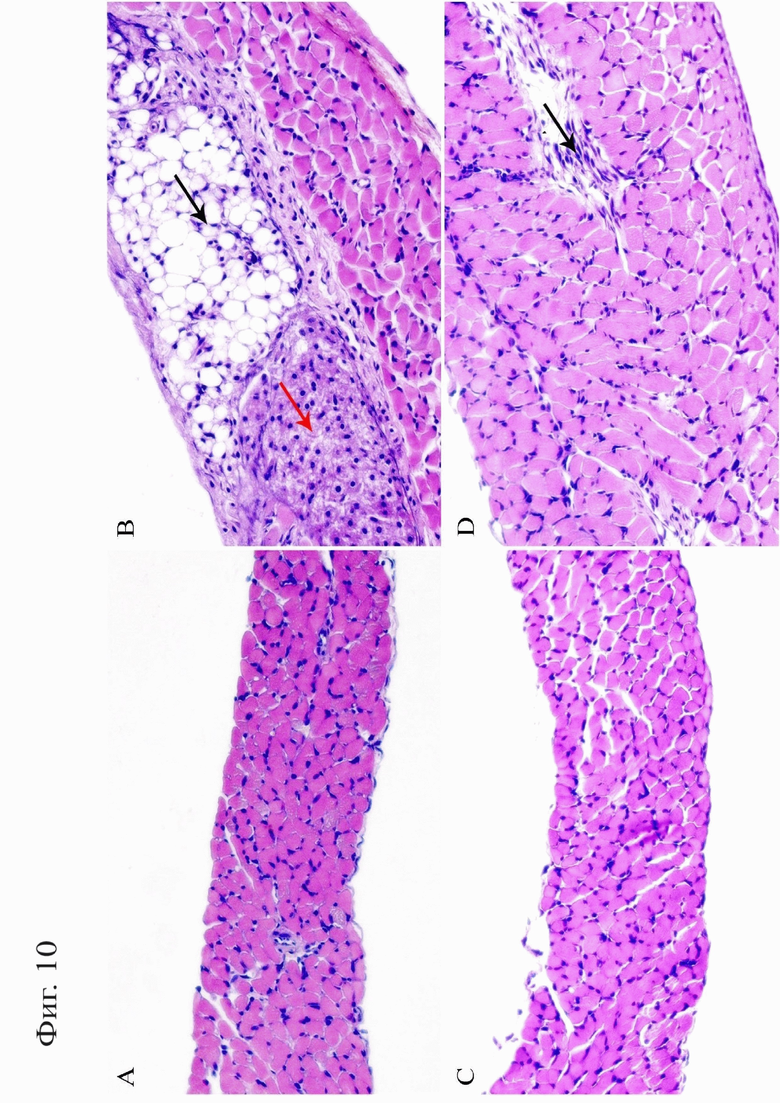
Фигура 10. Поперечные срезы диафрагмы, окраска гематоксилином и эозином, 10х. А, С - диафрагма мыши дикого типа. B - диафрагма мыши линии N2. D - диафрагма мыши линии N1.
Фигура 11. Продольные срезы сердца, окраска железным гематоксилином по Рего, 10х. А, С - миокард мыши дикого типа. B - миокард мыши линии N2. D - миокард мыши линии N1.
Осуществление изобретения
В описании данного изобретения, если не определено отдельно, технические и научные термины имеют стандартные значения, общепринятые в научной и технической литературе. В том числе, сокращение DMD используется для обозначения человеческого гена дистрофина, а сокращение Dmd для обозначения гена дистрофина мыши Mus musculus.
Специалисту в данной области техники должно быть понятно, что термины «гидовая РНК», «одиночная гидовая РНК», «сгРНК», «огРНК», «гРНК» и «sgRNA» являются взаимозаменяемыми и имеют одни и те же качества и значение.
Способ осуществляется следующим образом:
1. Получение генетических конструкций. Для введения выбранной мутации была подобрана с помощью онлайн инструмента http://crispor.tefor.net/ сгРНК, ПАМ-сайт которой находится в 51-м экзоне гена дистрофина мыши Dmd. Выбранная сгРНК имела последовательность 5'-TACTCTAGTGACACAATCTG/TGG-3' (SEQ ID NO: 2). Выбранная сгРНК была заклонирована в плазмидный вектор pSK-T7 (последовательностью SEQ ID NO: 1), предназначенный для in vitro синтеза сгРНК. Последовательность сгРНК была синтезирована с использованием двух олигонуклеотидов: sgDmd51-f олигонуклеотида для синтеза последовательности сгРНК последовательностью 5'-taggTACTCTAGTGACACAATCTG-3' (SEQ ID NO: 5) и sgDmd51-r олигонуклеотида для синтеза последовательности сгРНК последовательностью 5'-aaacCAGATTGTGTCACTAGAGTA-3' (SEQ ID NO: 6).
Разрезание вектора производили с использованием рестриктазы BbsI-HF (New England Biolabs), затем дефосфорилировали вектор с помощью фермента FastAP (Thermofisher Scientific), и очищали в агарозном геле и последующим выделением с помощью набора Monarch DNA Gel Extraction Kit. Олигонуклеотиды для последовательности сгРНК были заказаны с таким расчетом, чтобы их некомплиментарные концы образовали липкие концы для клонирования в вектор pSK-T7. Олигонуклеотиды фосфорилировали с использованием фермента PNK kinase (Thermofisher Scientific), отжигали друг с другом и лигировали с приготовленным ранее вектором и использованием фермента T4 DNA ligase (Thermofisher Scientific, США). Лигазную смесь трансформировали в компетентные клетки штамма XL1blue (Евроген). Скрининг колоний осуществляли с использованием ПЦР-наборов компании Евроген, прямого праймера для синтеза сгРНК (sgDmd51-f олигонуклеотида для синтеза последовательности сгРНК последовательностью 5'-taggTACTCTAGTGACACAATCTG-3' (SEQ ID NO: 5)) и обратного праймера к вектору (pSK T7 праймера для проверки конструкции и для амплификации фрагмента, используемого для in vitro синтеза гидовой РНК, последовательностью 5'-GCAAAAAAGCACCGACTC-3' (SEQ ID NO: 7)). По две колонии, содержащие сгРНК, наращивали в ночной культуре и выделяли с помощью набора Monarch Plasmid Miniprep Kit. Анализ полученных плазмид проводили методом секвенирования по Сэнгеру. Секвенирование полученных плазмид осуществляли с использованием pSK T7 праймера для проверки конструкции и для амплификации фрагмента, используемого для in vitro синтеза гидовой РНК, последовательностью 5'-GCAAAAAAGCACCGACTC-3' (SEQ ID NO: 7) в компании Евроген. Анализ электрофореграмм (сиквенсов) проводили с использованием программного обеспечения SnapGene.
2. Проверенную конструкцию использовали для in vitro синтеза гидовой РНК. В качестве матрицы для синтеза использовали ПЦР-фрагмент, полученный с использованием плазмиды из п.1 в качестве матрицы, двух праймеров:
pSK T7 праймера для проверки конструкции и для амплификации фрагмента, используемого для in vitro синтеза гидовой РНК, последовательностью 5'-GCAAAAAAGCACCGACTC-3' (SEQ ID NO: 7)
и M13 праймера для амплификации фрагмента, используемого для in vitro синтеза гидовой РНК, последовательностью 5'-GTTGTAAAACGACGGCCAGTG-3' (SEQ ID NO: 8),
а также высокоточной полимеразы Platinum SuperFi (Thermo Scientific). После амплификации 2 мкл реакционной смеси наносили на 1,8% агарозный гель для оценки полученного продукта (Фигура 1).
Далее полученный ПЦР-фрагмент очищали из реакционной смеси путем переосаждения с использованием 3М ацетата натрия, изопропанола и 70% этанола. Очищенный фрагмент использовали для in vitro синтеза гидовой РНК с применением набора HiScribe® T7 High Yield RNA Synthesis Kit (New England Biolabs). После проведения in vitro синтеза гидовой РНК реакционную смесь обрабатывали ферментом DNase I (New England Biolabs) с целью элиминации ДНК. Полученную гидовую РНК также очищали путем переосаждения и оценивали методом электрофореза (Фигура 2).
Далее проводили in vitro синтез РНК Cas9. В качестве матрицы для синтеза использовали плазмиду pET28a/Cas9-Cys (SEQ ID NO: 9), предварительно обработанную ферментом XhoI (Thermo Scientific). Полученный фрагмент очищали из реакционной смеси путем переосаждения с использованием 3М ацетата натрия, изопропанола и 70% этанола. Очищенный фрагмент использовали для in vitro синтеза РНК Cas9 с применением набора HiScribe™ T7 для синтеза мРНК с кэпированием ARCA, с полиаденилированием 3’-конца (New England Biolabs). После проведения in vitro синтеза реакционную смесь обрабатывали ферментом DNase I (New England Biolabs) с целью элиминации ДНК. Полученную РНК также очищали путем переосаждения и оценивали методом электрофореза (Фигура 3).
3. Получение яйцеклеток мышей. Яйцеклетки для микроинъекции получали методом индукции суперовуляции. Для этого неполовозрелым самкам гибридам F1(CBA×C57BL/6) весом 12-13 г внутрибрюшинно вводили 8 ед. гонадотропина сыворотки жеребых кобыл (ГСЖК) и через 48 час - 8 ед. хорионического гонадотропина человека (ХгЧ). После такой обработки самок ссаживали с самцами-производителями F1(CBA×C57BL/6). Факт спаривания констатировали на следующее утро по наличию копулятивной пробки.
Схема индукции суперовуляции: 12:00 - ГСЖК, через 48 ч. - ХгЧ. В 17:00 этого же дня - подсадка к самцам-производителям. Отбор доноров производили на следующий день в 9:00. Световой режим в виварии был установлен с 7:00 до 19:00.
Отобранных самок-доноров умерщвляли, извлекали яйцеводы, затем вымывали яйцеклетки в среде HEPES-KSOM с добавлением гиалуронидазы. Процедуру проводили под бинокуляром (Zeiss Stemi DV4) с увеличением в 32 раза. Для вымывания яйцеклеток использовали стеклянные капилляры с внутренним диаметром примерно 100 мкм, изготовленные на пуллере Narishige PC-10 (Япония) и микрокузнице Narishige MF-900 (Япония).
4. Микроинъекции. Полученные зиготы культивировали в течение двух часов при температуре 37 °C и 5% СО2 в капле среды KSOM под минеральным маслом (Sigma, США), затем помещали в микроинъекционную камеру. Микроинъекции проводили в среде HEPES-KSOM под микроскопом Zeiss Axiovert 200М при увеличении в 400–600 раз, используя микроманипуляторы Narishige. Для изготовления игл для микроинъекций использовали пуллер Sutter instrument Со Р-97 (США), для изготовления удерживающей пипетки использовали пуллер Narishige PC-10 и микрокузницу Narishige MF-900.
После окончания микроинъекций выжившие зиготы переносили в каплю среды KSOM под минеральное масло (Sigma) и культивировали в течение 1 часа для выявления жизнеспособных эмбрионов.
5. Получение самок-реципиентов. Самок-реципиентов яйцеклеток получали следующим образом: половозрелых самок F1(CBA×C57BL/6) весом не менее 24 г ссаживали с вазэктомированными самцами той же линии. Через 18 часов псевдобеременных реципиентов отбирали по наличию копулятивных пробок. Выжившие после микроинъекции зиготы трансплантировали в яйцеводы псевдобеременной самки. Одной псевдобеременной самке пересаживали 10-15 эмбрионов. Операцию проводили под наркозом (смесь золетила и рометара, вводился внутрибрюшинно).
6. Операция вазэктомирования. Операцию вазэктомирования проводили заранее под наркозом (смесь золетила и рометара, вводился внутрибрюшинно). Через надрез в коже и брюшной стенке вытягивали из брюшной полости семенник, придатки семенника и семявыносящий проток, после чего раскаленным пинцетом разрушали семявыносящий проток. Органы возвращали в брюшную полость, и повторяли всю процедуру на другом семявыносящем протоке. На завершающем этапе на брюшную стенку и на кожу накладывали швы с последующей антисептической обработкой операционного поля.
7. Получение новорожденных мышей. На 19-й день после пересадки микроинъецированных эмбрионов реципиента умерщвляли путём цервикальной дислокации и проводили кесарево сечение, после чего выживших детёнышей помещали к заранее подготовленной кормилице. Через 8–14 дней после рождения у мышат брали образец ткани, выделяли ДНК и анализировали наличие генетических модификаций.
8. Анализ наличия генетических модификаций в геноме животных производили методом секвенирования ПЦР-фрагмента. ДНК для ПЦР выделяли из тканей по стандартному протоколу. Амплификацию фрагментов ДНК проводили с помощью набора HS-Taq (Евроген, Москва) в присутствии 500 нМ двух праймеров:
Dmd–check-f олигонуклеотида для генотипирования последовательностью 5'-TGTTCTCTGGTGTACTGCCT-3' (SEQ ID NO: 3)
и Dmd-check-r олигонуклеотида для генотипирования последовательностью 5'-TGTTCACAGAGAAAAGGTAGCC-3' (SEQ ID NO: 4) -
в следующих режимах: денатурация 95 °С - 3 мин; далее следовало 35 циклов: 95 °С - 40 с, 58 °С - 40 с; 72 °С - 40 с; и финальная элонгация 72 °С - 2 мин. После прохождения реакции ПЦР-смесь очищали в 1,5% агарозном геле, полученный продукт выделяли набором Monarch DNA Gel Extraction Kit (New England Biolabs, США) и секвенировали в компании Евроген и использованием праймера – Dmd–check-f олигонуклеотида для генотипирования последовательностью 5'-TGTTCTCTGGTGTACTGCCT-3' (SEQ ID NO: 3). Полученные сиквенсы можно подразделить на три категории: сиквенсы дикого типа, последовательность в которых соответствует дикому типу, двоящиеся сиквенсы, соответствующие мозаичным животным F0 или гетерозиготным самкам следующих поколений, и сиквенсы, на которых присутствует только мутантный вариант ДНК, соответствующие трансгенным самцам и гомозиготным трансгенным самкам.
9. Определение генетически модифицированных животных в поколениях, начиная с F1, осуществляли с помощью ПЦР в реальном времени. Для этого применялись флуоресцентно меченные зонды с модифицированными замкнутыми нуклеотидами (LNA, locked nucleic acid, отмечены «[+A]» и «[+C]»), с ковалентно пришитой к 5'-концу последовательности молекулой флуоресцентного красителя 5(6)-карбоксифлуоресцеина (FAM) или флуорофора родамина Х/родамина 101 (ROX) и ковалентно пришитой к 3'-концу последовательности молекулой гасителя флуоресценции BHQ1 (Black Hole Quencher-1) или BHQ2 (Black Hole Quencher-2):
FAM-меченный зонд для определения аллели дикого варианта последовательностью 5'-(FAM)-AGTGACACA[+A]T[+C]TGTGGTTAC-(BHQ1)-3' (SEQ ID NO: 10);
ROX-меченный зонд для определения аллели “insT” последовательностью
5'-(ROX)-AGTGACACA[+A]TT[+C]TGTGGTTACTA-(BHQ2)-3' (SEQ ID NO: 11);
ROX-меченный зонд для определения аллели “insG” последовательностью 5'-(ROX)-TGACACA[+A]TG[+C]TGTGGTTACT-(BHQ2)-3' (SEQ ID NO: 12)
(описано в Примере 4)
и пара праймеров:
Dmd51 rt_gt f олигонуклеотид для ПЦР в реальном времени последовательностью 5'-AACACTAGCTGCCAGTCAGAC-3' (SEQ ID NO: 13);
Dmd51 rt_gt r олигонуклеотид для ПЦР в реальном времени последовательностью 5'-GTAAGTTCTGTCCAAGCTCGG-3' (SEQ ID NO: 14).
Пример 1. Основателем линии N1 стала самка “448”, с инсерцией нуклеотида T (insT). На фигуре 4 приведена хроматограмма сиквенса в районе PAM-сайта, а также реконструкция инсерции. Эта линия характеризуется нормальной продолжительностью жизни и менее выраженным фенотипом.
Пример 2. Основателем линии N2 стал самец 740 с инсерцией нуклеотида G (insG). На фигуре 5 приведена хроматограмма сиквенса в районе PAM-сайта, а также реконструкция инсерции. Эта линия характеризуется выраженным моторным фенотипом среди самцов, а также сокращенной продолжительностью жизни самцов.
Пример 3. Предсказание белковой последовательности мутантных вариантов. На основе электрофореграмм, полученных в результате секвенирования по Сэнгеру, восстанавливали последовательности аллелей модифицированного локуса. По данным последовательностям предсказывали последовательности вариантов продукта модифицированного гена дистрофина. Глобальное выравнивание производили методом Нидлмана-Вунша (Фиг. 6).
Инсерция дополнительного нуклеотида (insT или insG) в кодирующую последовательность гена дистрофина мыши (длина нативного белка - 3678 аминокислот), должна приводить к образованию двух укороченных вариантов. Мутация изменяет белок, начиная с 2441-й аминокислоты (АК). Оба варианта приводят к укорочению белка на 1233 АК - происходит терминация биосинтеза через три АК после мутировавшей.
В варианте insT происходит замена p.S2441F, в варианте insG - p.S2441C. Далее - укороченный трипептид CGY.
Пример 4. Генотипирование мышей линий N1 и N2 с помощью ПЦР в реальном времени. Определение генотипа (генотипирование) особей начиная с поколения F1 осуществляли с помощью ПЦР в реальном времени с применением флуоресцентно меченных зондов (Фиг. 7, SEQ ID NO: 10, 11, 12). Зонды содержат замкнутые нуклеотиды, окружающие определяемую мутацию - это позволяет значительно увеличить чувствительность и точность метода.
Выделяли очищенную ДНК исследуемых особей. В стандартную ПЦР-смесь с Taq-полимеразой с горячим стартом (Евроген, Россия) и праймерами:
Dmd51 rt_gt f олигонуклеотидом для ПЦР в реальном времени последовательностью 5'-AACACTAGCTGCCAGTCAGAC-3' (SEQ ID NO: 13)
и Dmd51 rt_gt r олигонуклеотидом для ПЦР в реальном времени последовательностью 5'-GTAAGTTCTGTCCAAGCTCGG-3' (SEQ ID NO: 14) -
добавляли флуоресцентно меченные зонды с модифицированными замкнутыми нуклеотидами (LNA, locked nucleic acid, отмечены «[+A]» и «[+C]»), с ковалентно пришитой к 5'-концу последовательности молекулой флуоресцентного красителя 5(6)-карбоксифлуоресцеина (FAM) или флуорофора родамина Х/родамина 101 (ROX) и ковалентно пришитой к 3'-концу последовательности молекулой гасителя флуоресценции BHQ1 (Black Hole Quencher-1) или BHQ2 (Black Hole Quencher-2). Для генотипирования животных линии N1 применяли зонды:
FAM-меченный зонд для определения аллели дикого варианта последовательностью 5'-(FAM)-AGTGACACA[+A]T[+C]TGTGGTTAC-(BHQ1)-3' (SEQ ID NO: 10);
ROX-меченный зонд для определения аллели “insT” последовательностью
5'-(ROX)-AGTGACACA[+A]TT[+C]TGTGGTTACTA-(BHQ2)-3' (SEQ ID NO: 11);
для генотипирования животных линии N2 применяли зонды:
FAM-меченный зонд для определения аллели дикого варианта последовательностью 5'-(FAM)-AGTGACACA[+A]T[+C]TGTGGTTAC-(BHQ1)-3' (SEQ ID NO: 10);
ROX-меченный зонд для определения аллели “insG” последовательностью 5'-(ROX)-TGACACA[+A]TG[+C]TGTGGTTACT-(BHQ2)-3' (SEQ ID NO: 12).
Проводили 40 циклов ПЦР с очищенной ДНК, по завершении дискриминировали аллели на основе интенсивности флуоресценции соответствующих зондов в смесях (Фиг. 8).
Система позволяет различить гомозиготные и гетерозиготные варианты генотипа. Поскольку ген дистрофина расположен в X-хромосоме, у самцов возможны только дикий и модифицированный генотипы, но не гетерозиготный.
Пример 5. Гистологический анализ
1. Забор образцов и фиксация. Некропсию и забор материала для гистологического исследования проводили согласно рекомендациям гистологического атласа A Practical Guide to the Histology of the Mouse (Cheryl L. Scudamore. A practical guide to the histology of the mouse. 2013. Print ISBN:9781119941200. Online ISBN:9781118789568. doi:10.1002/9781118789568). Для исследования забирали следующие органы: фрагмент икроножной мышцы, фрагмент межреберных мышц, диафрагму, сердце. Образцы помещали в гистологические кассеты (Биовитрум, Россия), фиксировали в 10% нейтральном забуференном формалине (Медикс, Россия) в течение суток.
2. Проводка. Фиксированные образцы отмывали от формалина под проточной водопроводной водой в течение одного часа. Далее осуществляли проводку по батарее дегидратанта (Медикс, Россия) восходящей концентрации (70%, 80%, 90%, 100%), проводили через промежуточные смеси №1 и №2 (Медикс, Россия) и минеральное масло (Медикс, Россия).
3. Заливка. Для заливки использовали парафин гистологический (Медикс, Россия) и парафин гистологический с воском (Медикс, Россия). Заливали в заливочные формы 7х7х6мм (Diapath, Италия). Проводку и заливку осуществляли согласно инструкциям производителя. Формы остужали до температуры 4°С, вынимали готовый блок, крепили на основание гистологической кассеты.
4. Получение срезов. Срезы толщиной 5 мкм получали на ротационном полуавтоматическом микротоме RMD-3000 (МТП, Россия) с использованием одноразовых лезвий Feather R35 (Япония). Полученные срезы расправляли на водяной бане “Слайдбаня-30/60” (КБ Техком, Россия), помещали на предметные стекла (CITOTEST, Китай), покрытые адгезивной жидкостью (Биовитрум, Россия). Стекла сушили не менее двух суток при комнатной температуре.
5. Окрашивание. Срезы депарафинировали при помощи депарафинирующего раствора (Медикс, Россия), проводили по батарее изопропанола (ЭКОС-1, Россия) нисходящей концентрации (100%, 90%, 70%) и помещали в дистиллированную воду. Срезы окрашивали гематоксилином Майера и эозином (Биовитрум, Россия) и железным гематоксилином Рего согласно инструкциям производителя, дегидратировали в изопропаноле (70%, 90%, 100%), просветляли в ксилоле (ЭКОС-1, Россия). Заключали под покровное стекло в монтирующую среду Витрогель (ЭргоПродакшн, Россия).
Микрофото получали при помощи микроскопа Nikon Eclipse Ti (Япония) с объективом Nikon Plan Fluor 10x/30 OFN25 Ph1 DL (Япония) и камеры E3ISPM21000KPA Microscope Camera (Sony, Япония).
Согласно данным литературы (Zweyer M, Sabir H, Dowling P, Gargan S, Murphy S, Swandulla D, Ohlendieck K. Histopathology of Duchenne muscular dystrophy in correlation with changes in proteomic biomarkers. Histol Histopathol. 2022 Feb;37(2):101-116. doi: 10.14670/HH-18-403. Epub 2021 Dec 7. PMID: 34873679.; Dubuisson N, Versele R, Planchon C, Selvais KM, Noel L, Michel Abou-Samra M, Davis-López de Carrizosa MA. Histological methods to assess skeletal muscle degeneration and regeneration in Duchenne muscular dystrophy. Int J Mol Sci. 2022 Dec 16;23(24):16080. doi: 10.3390/ijms232416080. PMID: 36555721.) к основным изменениям в мышечной ткани при миодистрофии Дюшенна относятся: вариабельность размеров миофибрилл, увеличение числа ядер, наличие волокон с центральной нуклеацией, наличие областей некроза и регенерации, наличие фрагментов иммунного воспаления. На поздних стадиях отмечают обширные области фиброза и замещение мышечных клеток адипоцитами.
Гистологический анализ срезов скелетных мышц на примере икроножной мышцы показал наличие у мышей линии N2 аномальной вариабельности диаметра мышечных волокон за счет большого количества гипертрофированных и атрофированных мышечных волокон и большое количество волокон с центральной нуклеацией и волокон с умеренным увеличением количества ядер (Фиг. 9, C). Замещения мышечных волокон жировой тканью не было обнаружено, однако в поле зрения присутствовали множественные очаги некроза с характерными фрагментами иммунного воспаления (Фиг. 9, С, черные стрелки). У самцов линии N1 также было выявлено наличие волокон с центральной нуклеацией и умеренная вариабельность диаметра мышечных волокон, однако областей некроза и иммунного воспаления не было обнаружено (Фиг. 9, E). Гистологический анализ межреберных мышц самцов обеих линий показал наличие очагов некроза на стадии миофагоцитоза (Фиг. 9, D, F, черные стрелки) и умеренную вариабельность диаметра мышечных волокон. Полученные данные соответствуют гистологической картине патологии мышечной ткани при миодистрофии Дюшенна.
Гистологический анализ диафрагмы показал разрастание перимизия, увеличение толщины диафрагмы, наличие обширных очагов замещения мышечных волокон жировой тканью (Фиг. 10, B, черные стрелки) и значительных очагов фиброза (Фиг. 10, B, красные стрелки) у самцов линии N2. У самцов линии N1 толщина диафрагмы также была увеличена и присутствовали локальные очаги фиброза (Фиг. 10, D, черные стрелки).
Наличие очагов жирового замещения в диафрагме является одним из гистологических признаков поздних стадий миодистрофии Дюшенна. На поздних стадиях миодистрофии патологический процесс становится системным и затрагивает другие органы, в частности сердце (Mavrogeni S, Markousis-Mavrogenis G, Papavasiliou A, Kolovou G. Cardiac involvement in Duchenne and Becker muscular dystrophy. World J Cardiol 2015 July 26; 7(7): 410-414. doi: 10.4330/wjc.v7.i7.410. PMID: 26225202; PMCID: PMC4513493.). Для исследования возможных патологий миокарда были исследованы сердца самцов линий N2 и N1. Железный гематоксилин по Рего был выбран с целью выявления ранних повреждений и некроза миокарда, однако никаких патологий в строении миокарда не было обнаружено (Фиг. 11).
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Duchenne
mouse.xml" softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2023-10-18">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>no</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт биологии гена Российской академии
наук </ApplicantName>
<ApplicantNameLatin>Institute of Gene Biology Russian Academy of
Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ получения модели для
изучения миодистрофии Дюшенна и вариантов ее терапии</InventionTitle>
<SequenceTotalQuantity>14</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>2543</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2543</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcgccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtt
tttccataggctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccg
acaggactataaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttccgaccctgc
cgcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctttctcatagctcacgctgtag
gtatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgac
cgctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgccactggcag
cagccactggtaacaggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcc
taactacggctacactagaaggacagtatttggtatctgcgctctgctgaagccagttaccttcggaaaa
agagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagc
agattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttctacggggtctgacgctcagtg
gaacgaaaactcacgttaagggattttggtcatgagattatcaaaaaggatcttcacctagatcctttta
aattaaaaatgaagttttaaatcaatctaaagtatatatgagtaaacttggtctgacagttaccaatgct
taatcagtgaggcacctatctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgt
gtagataactacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacgc
tcaccggctccagatttatcagcaataaaccagccagccggaagggccgagcgcagaagtggtcctgcaa
ctttatccgcctccatccagtctattaattgttgccgggaagctagagtaagtagttcgccagttaatag
tttgcgcaacgttgttgccattgctacaggcatcgtggtgtcacgctcgtcgtttggtatggcttcattc
agctccggttcccaacgatcaaggcgagttacatgatcccccatgttgtgcaaaaaagcggttagctcct
tcggtcctccgatcgttgtcagaagtaagttggccgcagtgttatcactcatggttatggcagcactgca
taattctcttactgtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattc
tgagaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacgggataataccgcgccacata
gcagaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaactctcaaggatcttaccgct
gttgagatccagttcgatgtaacccactcgtgcacccaactgatcttcagcatcttttactttcaccagc
gtttctgggtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataagggcgacacggaaatgtt
gaatactcatactcttcctttttcaatattattgaagcatttatcagggttattgtctcatgagcggata
catatttgaatgtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgccacct
aaattgtaagcgttaatattttgttaaaattcgcgttaaatttttgttaaatcagctcattttttaacca
ataggccgaaatcggcaaaatcccttataaatcaaaagaatagaccgagatagggttgagtgttgttcca
gtttggaacaagagtccactattaaagaacgtggactccaacgtcaaagggcgaaaaaccgtctatcagg
gcgatggcccactacgtgaaccatcaccctaatcaagttttttggggtcgaggtgccgtaaagcactaaa
tcggaaccctaaagggagcccccgatttagagcttgacggggaaagccggcgaacgtggcgagaaaggaa
gggaagaaagcgaaaggagcgggcgctagggcgctggcaagtgtagcggtcacgctgcgcgtaaccacca
cacccgccgcgcttaatgcgccgctacagggcgcgtcccattcgccattcaggctgcgcaactgttggga
agggcgatcggtgcgggcctcttcgctattacgccagctggcgaaagggggatgtgctgcaaggcgatta
agttgggtaacgccagggttttcccagtcacgacgttgtaaaacgacggccagtgaattgtaatacgact
cactatagggcgtcttctagaagacctgttttagagctagaaatagcaagttaaaataaggctagtccgt
tatcaacttgaaaaagtggcaccgagtcggtgcttttttgccgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tactctagtgacacaatctgtgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgttctctggtgtactgcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgttcacagagaaaaggtagcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taggtactctagtgacacaatctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaccagattgtgtcactagagta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcaaaaaagcaccgactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttgtaaaacgacggccagtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>9546</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9546</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgtagaggatcgagatctcgatcccgcgaaattaatacgactcactat
aggggaattgtgagcggataacaattcccctctagaaataattttgtttaactttaagaaggagatatac
catgggcagcagccatcatcatcatcatcacagcagcggcctggtgccgcgcggcagccatatggctagc
atgactggtggacagcaaatgggtcgcggatccgaattcgagctccgtcgacaagcttgcggccgcatgg
acaagaagtacagcatcggcctggacatcggtaccaacagcgtgggctgggccgtgatcaccgacgagta
caaggtgcccagcaagaagttcaaggtgctgggcaacaccgaccgccacagcatcaagaagaacctgatc
ggcgccctgctgttcgacagcggcgagaccgccgaggccacccgcctgaagcgcaccgcccgccgccgct
acacccgccgcaagaaccgcatctgctacctgcaggagatcttcagcaacgagatggccaaggtggacga
cagcttcttccaccgcctggaggagagcttcctggtggaggaggacaagaagcacgagcgccaccccatc
ttcggcaacatcgtggacgaggtggcctaccacgagaagtaccccaccatctaccacctgcgcaagaagc
tggtggacagcaccgacaaggccgacctgcgcctgatctacctggccctggcccacatgatcaagttccg
cggccacttcctgatcgagggcgacctgaaccccgacaacagcgacgtggacaagctgttcatccagctg
gtgcagacctacaaccagctgttcgaggagaaccccatcaacgccagcggcgtggacgccaaggccatcc
tgagcgcccgcctgagcaagagccgccgcctggagaacctgatcgcccagctgcccggcgagaagaagaa
cggcctgttcggcaacctgatcgccctgagcctgggcctgacccccaacttcaagagcaacttcgacctg
gccgaggacgccaagctgcagctgagcaaggacacctacgacgacgacctggacaacctgctggcccaga
tcggcgaccagtacgccgacctgttcctggccgccaagaacctgagcgacgccatcctgctgagcgacat
cctgcgcgtgaacaccgagatcaccaaggcccccctgagcgccagcatgatcaagcgctacgacgagcac
caccaggacctgaccctgctgaaggccctggtgcgccagcagctgcccgagaagtacaaggagatcttct
tcgaccagagcaagaacggctacgccggctacatcgacggcggcgccagccaggaggagttctacaagtt
catcaagcccatcctggagaagatggacggcaccgaggagctgctggtgaagctgaaccgcgaggacctg
ctgcgcaagcagcgcaccttcgacaacggcagcatcccccaccagatccacctgggcgagctgcacgcca
tcctgcgccgccaggaggacttctaccccttcctgaaggacaaccgcgagaagatcgagaagatcctgac
cttccgcatcccctactacgtgggccccctggcccgcggcaacagccgcttcgcctggatgacccgcaag
agcgaggagaccatcaccccctggaacttcgaggaggtggtggacaagggcgccagcgcccagagcttca
tcgagcgcatgaccaacttcgacaagaacctgcccaacgagaaggtgctgcccaagcacagcctgctgta
cgagtacttcaccgtgtacaacgagctgaccaaggtgaagtacgtgaccgagggcatgcgcaagcccgcc
ttcctgagcggcgagcagaagaaggccatcgtggacctgctgttcaagaccaaccgcaaggtgaccgtga
agcagctgaaggaggactacttcaagaagatcgagtgcttcgacagcgtggagatcagcggcgtggagga
ccgcttcaacgccagcctgggcacctaccacgacctgctgaagatcatcaaggacaaggacttcctggac
aacgaggagaacgaggacatcctggaggacatcgtgctgaccctgaccctgttcgaggaccgcgagatga
tcgaggagcgcctgaagacctacgcccacctgttcgacgacaaggtgatgaagcagctgaagcgccgccg
ctacaccggctggggccgcctgagccgcaagcttatcaacggcatccgcgacaagcagagcggcaagacc
atcctggacttcctgaagagcgacggcttcgccaaccgcaacttcatgcagctgatccacgacgacagcc
tgaccttcaaggaggacatccagaaggcccaggtgagcggccagggcgacagcctgcacgagcacatcgc
caacctggccggcagccccgccatcaagaagggcatcctgcagaccgtgaaggtggtggacgagctggtg
aaggtgatgggccgccacaagcccgagaacatcgtgatcgagatggcccgcgagaaccagaccacccaga
agggccagaagaacagccgcgagcgcatgaagcgcatcgaggagggcatcaaggagctgggcagccagat
cctgaaggagcaccccgtggagaacacccagctgcagaacgagaagctgtacctgtactacctgcagaac
ggccgcgacatgtacgtggaccaggagctggacatcaaccgcctgagcgactacgacgtggaccacatcg
tgccccagagcttcctgaaggacgacagcatcgacaacaaggtgctgacccgcagcgacaagaaccgcgg
caagagcgacaacgtgcccagcgaggaggtggtgaagaagatgaagaactactggcgccagctgctgaac
gccaagctgatcacccagcgcaagttcgacaacctgaccaaggccgagcgcggcggcctgagcgagctgg
acaaggccggcttcatcaagcgccagctggtggagacccgccagatcaccaagcacgtggcccagatcct
ggacagccgcatgaacaccaagtacgacgagaacgacaagctgatccgcgaggtgaaggtgatcaccctg
aagagcaagctggtgagcgacttccgcaaggacttccagttctacaaggtgcgcgagatcaacaactacc
accacgcccacgacgcctacctgaacgccgtggtgggcaccgccctgatcaagaagtaccccaagctgga
gagcgagttcgtgtacggcgactacaaggtgtacgacgtgcgcaagatgatcgccaagagcgagcaggag
atcggcaaggccaccgccaagtacttcttctacagcaacatcatgaacttcttcaagaccgagatcaccc
tggccaacggcgagatccgcaagcgccccctgatcgagaccaacggcgagaccggcgagatcgtgtggga
caagggccgcgacttcgccaccgtgcgcaaggtgctgagcatgccccaggtgaacatcgtgaagaagacc
gaggtgcagaccggcggcttcagcaaggagagcatcctgcccaagcgcaacagcgacaagctgatcgccc
gcaagaaggactgggaccccaagaagtacggcggcttcgacagccccaccgtggcctacagcgtgctggt
ggtggccaaggtggagaagggcaagagcaagaagctgaagagcgtgaaggagctgctgggcatcaccatc
atggagcgcagcagcttcgagaagaaccccatcgacttcctggaggccaagggctacaaggaggtgaaga
aggacctgatcatcaagctgcccaagtacagcctgttcgagctggagaacggccgcaagcgcatgctggc
cagcgccggcgagctgcagaagggcaacgagctggccctgcccagcaagtacgtgaacttcctgtacctg
gccagccactacgagaagctgaagggcagccccgaggacaacgagcagaagcagctgttcgtggagcagc
acaagcactacctggacgagatcatcgagcagatcagcgagttcagcaagcgcgtgatcctggccgacgc
caacctggacaaggtgctgagcgcctacaacaagcaccgcgacaagcccatccgcgagcaggccgagaac
atcatccacctgttcaccctgaccaacctgggcgcccccgccgccttcaagtacttcgacaccaccatcg
accgcaagcgctacaccagcaccaaggaggtgctggacgccaccctgatccaccagagcatcaccggtct
gtacgagacccgcatcgacctgagccagctgggcggcgacggcggctccggacctccaaagaaaaagaga
aaagtatacccctacgacgtgcccgactacgcctgttaagcggccgcactcgagcaccaccaccaccacc
actgagatccggctgctaacaaagcccgaaaggaagctgagttggctgctgccaccgctgagcaataact
agcataaccccttggggcctctaaacgggtcttgaggggttttttgctgaaaggaggaactatatccgga
ttggcgaatgggacgcgccctgtagcggcgcattaagcgcggcgggtgtggtggttacgcgcagcgtgac
cgctacacttgccagcgccctagcgcccgctcctttcgctttcttcccttcctttctcgccacgttcgcc
ggctttccccgtcaagctctaaatcgggggctccctttagggttccgatttagtgctttacggcacctcg
accccaaaaaacttgattagggtgatggttcacgtagtgggccatcgccctgatagacggtttttcgccc
tttgacgttggagtccacgttctttaatagtggactcttgttccaaactggaacaacactcaaccctatc
tcggtctattcttttgatttataagggattttgccgatttcggcctattggttaaaaaatgagctgattt
aacaaaaatttaacgcgaattttaacaaaatattaacgcttacaatttaggtggcacttttcggggaaat
gtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccgctcatgaattaattct
tagaaaaactcatcgagcatcaaatgaaactgcaatttattcatatcaggattatcaataccatattttt
gaaaaagccgtttctgtaatgaaggagaaaactcaccgaggcagttccataggatggcaagatcctggta
tcggtctgcgattccgactcgtccaacatcaatacaacctattaatttcccctcgtcaaaaataaggtta
tcaagtgagaaatcaccatgagtgacgactgaatccggtgagaatggcaaaagtttatgcatttctttcc
agacttgttcaacaggccagccattacgctcgtcatcaaaatcactcgcatcaaccaaaccgttattcat
tcgtgattgcgcctgagcgagacgaaatacgcgatcgctgttaaaaggacaattacaaacaggaatcgaa
tgcaaccggcgcaggaacactgccagcgcatcaacaatattttcacctgaatcaggatattcttctaata
cctggaatgctgttttcccggggatcgcagtggtgagtaaccatgcatcatcaggagtacggataaaatg
cttgatggtcggaagaggcataaattccgtcagccagtttagtctgaccatctcatctgtaacatcattg
gcaacgctacctttgccatgtttcagaaacaactctggcgcatcgggcttcccatacaatcgatagattg
tcgcacctgattgcccgacattatcgcgagcccatttatacccatataaatcagcatccatgttggaatt
taatcgcggcctagagcaagacgtttcccgttgaatatggctcataacaccccttgtattactgtttatg
taagcagacagttttattgttcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcagac
cccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaa
aaaaaccaccgctaccagcggtggtttgtttgccggatcaagagccaccaactctttttccgaaggtaac
tggcttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccaccacttcaag
aactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcgata
agtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacggg
gggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagcta
tgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacag
gagagcgcacgagggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacct
ctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcg
gcctttttacggttcctggccttttgctggccttttgctcacatgttctttcctgcgttatcccctgatt
ctgtggataaccgtattaccgcctttgagtgagctgataccgctcgccgcagccgaacgaccgagcgcag
cgagtcagtgagcgaggaagcggaagagcgcctgatgcggtattttctccttacgcatctgtgcggtatt
tcacaccgcaatggtgcactctcagtacaatctgctctgatgccgcatagttaagccagtatacactccg
ctatcgctacgtgactgggtcatggctgcgccccgacacccgccaacacccgctgacgcgccctgacggg
cttgtctgctcccggcatccgcttacagacaagctgtgaccgtctccgggagctgcatgtgtcagaggtt
ttcaccgtcatcaccgaaacgcgcgaggcagctgcggtaaagctcatcagcgtggtcgtgaagcgattca
cagatgtctgcctgttcatccgcgtccagctcgttgagtttctccagaagcgttaatgtctggcttctga
taaagcgggccatgttaagggcggttttttcctgtttggtcactgatgcctccgtgtaagggggatttct
gttcatgggggtaatgataccgatgaaacgagagaggatgctcacgatacgggttactgatgatgaacat
gcccggttactggaacgttgtgagggtaaacaactggcggtatggatgcggcgggaccagagaaaaatca
ctcagggtcaatgccagcgcttcgttaatacagatgtaggtgttccacagggtagccagcagcatcctgc
gatgcagatccggaacataatggtgcagggcgctgacttccgcgtttccagactttacgaaacacggaaa
ccgaagaccattcatgttgttgctcaggtcgcagacgttttgcagcagcagtcgcttcacgttcgctcgc
gtatcggtgattcattctgctaaccagtaaggcaaccccgccagcctagccgggtcctcaacgacaggag
cacgatcatgcgcacccgtggggccgccatgccggcgataatggcctgcttctcgccgaaacgtttggtg
gcgggaccagtgacgaaggcttgagcgagggcgtgcaagattccgaataccgcaagcgacaggccgatca
tcgtcgcgctccagcgaaagcggtcctcgccgaaaatgacccagagcgctgccggcacctgtcctacgag
ttgcatgataaagaagacagtcataagtgcggcgacgatagtcatgccccgcgcccaccggaaggagctg
actgggttgaaggctctcaagggcatcggtcgagatcccggtgcctaatgagtgagctaacttacattaa
ttgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagctgcattaatgaatcggcca
acgcgcggggagaggcggtttgcgtattgggcgccagggtggtttttcttttcaccagtgagacgggcaa
cagctgattgcccttcaccgcctggccctgagagagttgcagcaagcggtccacgctggtttgccccagc
aggcgaaaatcctgtttgatggtggttaacggcgggatataacatgagctgtcttcggtatcgtcgtatc
ccactaccgagatatccgcaccaacgcgcagcccggactcggtaatggcgcgcattgcgcccagcgccat
ctgatcgttggcaaccagcatcgcagtgggaacgatgccctcattcagcatttgcatggtttgttgaaaa
ccggacatggcactccagtcgccttcccgttccgctatcggctgaatttgattgcgagtgagatatttat
gccagccagccagacgcagacgcgccgagacagaacttaatgggcccgctaacagcgcgatttgctggtg
acccaatgcgaccagatgctccacgcccagtcgcgtaccgtcttcatgggagaaaataatactgttgatg
ggtgtctggtcagagacatcaagaaataacgccggaacattagtgcaggcagcttccacagcaatggcat
cctggtcatccagcggatagttaatgatcagcccactgacgcgttgcgcgagaagattgtgcaccgccgc
tttacaggcttcgacgccgcttcgttctaccatcgacaccaccacgctggcacccagttgatcggcgcga
gatttaatcgccgcgacaatttgcgacggcgcgtgcagggccagactggaggtggcaacgccaatcagca
acgactgtttgcccgccagttgttgtgccacgcggttgggaatgtaattcagctccgccatcgccgcttc
cactttttcccgcgttttcgcagaaacgtggctggcctggttcaccacgcgggaaacggtctgataagag
acaccggcatactctgcgacatcgtataacgttactggtttcacattcaccaccctgaattgactctctt
ccgggcgctatcatgccataccgcgaaaggttttgcgccattcgatggtgtccgggatctcgacgctctc
ccttatgcgactcctgcattaggaagcagcccagtagtaggttgaggccgttgagcaccgccgccgcaag
gaatggtgcatgcaaggagatggcgcccaacagtcccccggccacggggcctgccaccatacccacgccg
aaacaagcgctcatgagcccgaagtggcgagcccgatcttccccatcggtgatgtcggcgatataggcgc
cagcaaccgcacctgtggcgccggtgatgccggccacgatgcgtccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>5'UTR</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>FAM</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LNA (locked nucleic
acid)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>12</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LNA (locked nucleic
acid)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>3'UTR</INSDFeature_key>
<INSDFeature_location>>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q40">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>BHQ1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtgacacaatctgtggttac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>5'UTR</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q32">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ROX</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LNA (locked nucleic
acid)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LNA (locked nucleic
acid)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>3'UTR</INSDFeature_key>
<INSDFeature_location>>24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q39">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>BHQ2</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtgacacaattctgtggttacta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>5'UTR</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q35">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ROX</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LNA (locked nucleic
acid)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q37">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>LNA (locked nucleic
acid)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>3'UTR</INSDFeature_key>
<INSDFeature_location>>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q38">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>BHQ2</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgacacaatgctgtggttact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacactagctgccagtcagac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtaagttctgtccaagctcgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ получения генно-модифицированных лабораторных животных с нуль-аллелем гена P2rx3 | 2022 |
|
RU2805173C1 |
| Способ получения генно-модифицированных кроликов с нокаутом гена LEPR с помощью системы CRISPR/Cas9 | 2023 |
|
RU2836438C1 |
| Способ получения генно-модифицированных мышей, экспрессирующих миниген антитромбина III человека, с помощью микроинъекций TelN-линеаризованного фрагмента ДНК | 2022 |
|
RU2806568C1 |
| АНТИСМЫСЛОВЫЕ ОЛИГОНУКЛЕОТИДЫ, НАЦЕЛЕННЫЕ НА ГЕН CAV3.1, И ИХ ПРИМЕНЕНИЯ | 2022 |
|
RU2839721C2 |
| Система направленного изменения сплайсинга в гене MARK2 | 2023 |
|
RU2810907C1 |
| Способ получения нокаута гена CD209 в эмбрионах Bos taurus путем трансдукции зигот адено-ассоциированными вирусами, кодирующими saCas9 и соответствующую гидовую РНК | 2022 |
|
RU2800917C1 |
| ЛИНИЯ ГЕНЕТИЧЕСКИ МОДИФИЦИРОВАННЫХ МЫШЕЙ ДЛЯ МОДЕЛИРОВАНИЯ БОЛЕЗНИ АЛЬЦГЕЙМЕРА, СПОСОБ ЕЕ ПОЛУЧЕНИЯ И ЕЕ ПРИМЕНЕНИЕ, sgPHK И КОМБИНАЦИЯ crRNA/tracrRNA, ИСПОЛЬЗУЕМЫЕ В СПОСОБЕ, И НАБОР ПРАЙМЕРОВ | 2024 |
|
RU2835497C1 |
| Способ анализа соматических мутаций в генах IDH1 и IDH2 с использованием LNA-блокирующей ПЦР и гибридизации с биологическим микрочипом | 2024 |
|
RU2839291C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА GBSSI У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2817377C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА GBSSI У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2834229C1 |
Изобретение относится к области биотехнологии, в частности к плазмидной конструкции, полученной на основе вектора pSK-T7 и использованной для in vitro синтеза гидовой РНК против фрагмента последовательности 51-го экзона гена Dmd, а также к способу получения линии мышей, содержащих мутацию в 51-м экзоне. Изобретение позволяет создавать линии генетически модифицированных мышей для моделирования миодистрофии Дюшенна и разработки терапевтических подходов. 3 н.п. ф-лы, 11 ил., 5 пр.
1. Плазмидная конструкция для in vitro синтеза гидовой РНК, несущая вектор pSK-T7 последовательностью SEQ ID NO: 1 и кодирующая гидовую РНК последовательностью SEQ ID NO: 2 против соответствующего участка гена дистрофина мыши Dmd под контролем T7 промотора.
2. Способ получения конструкции по п.1, характеризующийся следующими стадиями:
a) биоинформатический анализ последовательности гена дистрофина мыши Dmd и подбор гидовой РНК в области экзона 51;
b) клонирование последовательности гидовой РНК в вектор pSK-T7 последовательностью SEQ ID NO: 1.
3. Способ получения линий мышей с мутациями в 51-ом экзоне гена дистрофина мыши Dmd, приводящими к сдвигу рамки считывания, характеризующийся следующими стадиями:
a) c помощью конструкции по п.1 осуществляют синтез in vitro гидовой РНК последовательностью SEQ ID NO: 2 против 51-го экзона гена дистрофина мыши Dmd, а также мРНК гена spCas9;
b) далее проводят микроинъекцию смеси гидовой РНК и мРНК spCas9 из пункта (a) в цитоплазму оплодотворенной яйцеклетки мыши гибрида первого поколения CBA×C57BL/6;
c) выявляют жизнеспособных зигот;
d) пересаживают выживших зигот из пункта (c) псевдобеременным самкам-реципиентам, имеющим копулятивную пробку после ссаживания с вазэктомированными самцами;
e) получают новорожденных мышей на 19 день после пересадки зигот из пункта (d);
f) проводят анализ наличия мутации через 8-14 дней после рождения мышей (e), путем амплификации целевого участка гена дистрофина мыши Dmd с двумя праймерами: Dmd-check-f олигонуклеотидом для генотипирования, имеющего последовательность 5'-TGTTCTCTGGTGTACTGCCT-3' (SEQ ID NO: 3) и Dmd-check-r олигонуклеотидом для генотипирования, имеющего последовательность 5'-TGTTCACAGAGAAAAGGTAGCC-3' (SEQ ID NO: 4) - с последующим секвенированием фрагмента;
g) отбирают мышей, несущих целевую мутацию;
h) проводят скрещивание мышей из пункта (g) с мышами дикого типа для получения самок-гетерозигот или мутантных самцов, несущих целевую мутацию.
| Способ получения мышиной модели для изучения синдрома Леша-Нихена путем внесения делеции p.Val8del в ген hprt1 | 2021 |
|
RU2768048C1 |
| Способ получения линии гуманизированных мышей, содержащих инсерцию 3974insT в гене mGrin2a (mice glutamate [NMDA] receptor subunit epsilon-1), приводящую к преждевременному прекращению трансляции белка grin2a | 2021 |
|
RU2764650C1 |
| WO 2021222327 A1, 04.11.2021 | |||
| WO 2017095967 A2, 08.06.2017 | |||
| XU L | |||
| et al | |||
| CRISPR-mediated Genome Editing Restores Dystrophin Expression and Function in mdx Mice, Mol Ther., 2016, Volume 24(3), pp | |||
| Прибор для механического вычерчивания аксонометрических проекции, симметрических фигур, обращенных изображений и для копирования чертежей | 1923 |
|
SU564A1 |
Авторы
Даты
2024-03-25—Публикация
2023-10-31—Подача