Изобретение относится к области молекулярной биологии и прогностических тестов и используется для определения вероятного этногеографического происхождения человека. Изобретение представляет собой набор синтетических олигонуклеотидных праймеров для проведения мультиплексной полимеразной цепной реакции (ПЦР). Изобретение дает возможность определить генотип по однонуклеотидным ДНК маркерам для последующего определения этногеографического происхождения человека из образцов геномной ДНК человека, в том числе в случае, когда ДНК в исследуемом образце находится в деградированном виде. Преимущество данного изобретения по сравнению с другими аналогами заключается в возможности проведения успешного анализа даже при использовании сильно деградировавших образцов ДНК.
Уровень техники
Системы для ДНК-идентификации популяционного и этногеографического происхождения человека необходимы как для криминалистических целей, так и широкого круга научных задач, таких, например, как подбор корректных групп индивидов при проведении исследований типа случай/контроль, определения происхождения древних индивидов из археологических раскопок. Для таких целей преимущественно используют ДНК-маркеры, характеризующиеся наибольшими различиями в частотах распределения аллелей между различными популяциями и этническими группами [1-3]. В судебных расследованиях такая информация о происхождении индивида, биологический материал которого изучается, может сыграть решающую роль [4, 5].
Для получения информации о происхождении индивида могут быть использованы четыре основных типа генетических маркеров: митохондриальная ДНК (мтДНК), маркеры Y-хромосомы, аутосомные короткие тандемные повторы (STR) и однонуклеотидные полиморфизмы (SNP). Гаплотипы Y-хромосомы и мтДНК уже показали свою эффективность при изучении миграции предков современных людей [6-8]. Однако при подобном анализе с использованием Y-хромосомных и мтДНК-маркеров абсолютно не учитывается информации основной части генома аутосомных. Полиморфные аутосомные STR-маркеры позволяют успешно отличить одного человека от другого (за исключением однояйцевых близнецов), однако они слабо применимы при анализе происхождения, так как большинство людей имеют одинаковые STR-аллели, а STR-маркеры характеризуются высокой скоростью мутирования. Кроме того, STR-аллели одного и того же размера могут иметь разное происхождение [9]. В свою очередь, аутосомные SNP-маркеры имеют относительно низкую скорость мутации; один и тот же SNP-аллель часто оказывается идентичен по происхождению у разных индивидов, а миллионы выявленных к настоящему времени SNP-маркеров в геноме человека доступны в публичных базах данных, например, в базе данных dbSNP, в проекте International НарМар [11] и 1000 Genomes [12] В связи с этим аутосомные SNP считаются в настоящее время лучшими кандидатами для определения этногеографического или популяционного происхождения индивида и ряд из них уже вошел в несколько разработанных для применения в судебной практике панелей SNP-маркеров [13-17]. Первые тесты в судебной генетической экспертизе, связанные с определением этногеографического происхождения индивида, были основаны на данных, полученных в рамках проекта Human Genome Mapping Project SNP Map [18], а также международного проекта НарМар [19]. Вскоре была опубликована первая панель SNP-маркеров, наиболее информативных для определения этногеографического происхождения индивида [20]. В идеальном случае такой маркер должен иметь один аллель, зафиксированный в одной популяции, и полностью отсутствующий в другой, однако большинство аллелей являются общими для популяций. Для отбора наиболее информативных маркеров были разработаны статистические критерии, включая абсолютные разности частот аллелей, F-статистику и информативность [21-23], а также проведено сравнение этих альтернативных методов [21, 24]. Наиболее перспективные уже были использованы для отбора информативных SNP для определения этнической принадлежности индивида [13, 17, 25]. Значительным достижением стала публикация почти 80 млн однонуклеотидных вариаций, идентифицированных у ~2.5 тыс.представителей 26 популяций в рамках проекта 1000 Геномов (http://www.internationalgenome.org). Все эти данные способствовали активной разработке наборов SNP-маркеров и появлению на рынке различных коммерческих наборов для определения этногеографического происхождения, как в общемировом масштабе, так и применительно к отдельным географическим регионам или странам [26-28]. За последнее десятилетие достижения в области геномных технологий, позволяющих проводить полногеномное генотипирование и секвенирование, позволило исследовать структуру мирового населения с помощью анализа маркеров по всему геному. С применением технологии параллельного секвенирования был разработан алгоритм, позволяющий на основе анализа 40000-130000 однонуклеотидных полиморфизмов установить страну происхождения для 87% индивидов мирового населения [29]. В последние время предложены новые методы, позволяющие на основе полногеномных данных установить происхождение конкретного индивида [30, 31]. Стоит отметить дополнительный вклад в генетику российских популяции полногеномного исследования Wong EH et al. (2017), раскрывающего исторические корни народов Сибири и Северо-Запада России [32]. Исследование популяций европейской территории России с помощью 166000 однонуклеотидных полиморфизмов по всему геному показало возможность разделения популяций этой области на основе генетических данных [33].
Среди коммерческие для наборов определения этнической принадлежности можно выделить Infinium Multi-Ethnic Global-8 Kit компании Illumina. Набор Infinium Multi-Ethnic Global-8 Kit компании Illumina представляет собой чип для генотипирования более 1 779 819 различных вариантов (SNP, VNTR, инсерции/делеции, структурные варианты, генеративные варианты) с общими и редкими признаками, которые могут быть использованы для различных целей, к примеру, в медицинских и популяционных исследованиях. Данный набор оптимизирован под различные популяционные группы. Также предлагаются наборы отдельно для испаноязычного и афро-американского происхождения (Product hifographic Infinium Multi-Ethnic AMR/AFR-8 Kit) и европейских, восточно- и южноазиатских популяций (Product Infographic Infinium Multi-Ethnic EUR/EAS/SAS-8 Kit). Однако, следует отметить, что данные наборы могут быть использованы только для научных исследований.
В то же время, следует отметить, что, ДНК-анализ в современной криминалистике сталкивается с целым рядом особенностей. Криминалистические образцы чаще всего попадают в руки экспертов далеко не сразу, и подвергаются воздействию эндогенных и экзогенных факторов, способствующих деградации целевой ДНК. Наибольшую часть исследуемых образцов ДНК составляют такие образцы ДНК из пятен крови различной давности, волос, костной ткани, потожировые отпечатки и т.д. Поэтому, в криминалистической практике достаточно часто встречаются случаи, когда для генетического анализа доступны лишь малые количества ДНК или ДНК в биологических материалах высоко деградировала. Такой тип генетического материала требует особого подхода для ДНК-анализа и интерпретирования полученных результатов экспертизы. Следовательно, в настоящее время стоит вопрос о необходимости разработки панели SNP-маркеров, которые были бы эффективны для решения такой существенной проблемы в работе с криминалистическими образцами, как малый размер ДНК-матрицы и малые количества образца. Также, следует отметить, что на сегодняшний день нет референсных панелей информативных SNP-маркеров для определения этногеографического происхождения индивидов из российский популяций, рекомендованных для судебно-медицинской экспертизы, так же как отсутствуют утвержденные критерии выбора таких маркеров.
Таким образом, техническая проблема, решаемая настоящим изобретением, заключается в расширении арсенала технических средств, которые используются для определения вероятного этногеографического происхождения индивидуумов на территории России при работе с криминалистическим образцами, в которых ДНК сильно деградирована.
Выбор набора маркеров
Авторами были выбраны 55 ДНК маркера, которые в настоящее время наиболее широко рекомендованные для определения этногеографического определения неизвестных индивидов [13]. Выбранные маркеры являются высокоинформативными однонуклеотидными полиморфизмами, частоты которых значительно варьируют в 73 протестированных популяциях, среди которых было 16 африканских; 24 азиатских; 18 европейских (в том числе русские из Архангельской области, русские из Вологодской области); 3 сибирские: коми, ханты и якуты; 5 тихоокеанских островных и 7 американских популяций. Выбранный набор маркеров хорошо оптимизирован под европейские популяции, но также, позволяет определять азиатские популяции, что имеет значение при ДНК-анализе криминалистических образцов индивидов на территории России, где представлено большое число и разнообразие этнических групп, как европейских, так и азиатских.
К выбранному набору ДНК-маркеров авторами были подобраны оригинальные олигонуклеотидные праймеры (табл. 1). таким образом, чтобы полученный ПНР-продукт имел минимальную длину, что позволило бы повысить результативность при исследовании деградированной ДНК. Оригинальные олигонуклеотидные праймеры были подобраны также с учетом того, чтобы можно было эффективно амплифицировать все целевые локусы в ходе одной мультиплексной ПЦР, чтобы маркеры могли быть детектированы методом NGS одновременно. Такой подход значительно уменьшает расход матричной ДНК образца, что принципиально важно работ с криминалистическим материалом, который часто бывает в доступности в ограниченных количествах.
В связи с этим, разработанные авторами оригинальные олигонуклеотидные последовательности праймеров соответствовали следующим критериям:
1. Разработанная пара олигонуклеотидов должна обеспечивать специфическую амплификацию конкретного участка генома человека, содержащего выбранный ДНК маркер
2. Размер амплифицированного фрагмента, содержащего выбранный маркер, должен быть менее 150 пар оснований.
3. На 3' конце последовательности олигонуклеотида не должно содержаться известных полиморфизмов, согласно базе данных gnomAD
4. Температуры отжига разработанных праймеров должны варьироваться от 57 до 60 градусов Цельсия
5. Все 110 разработанных олигонуклеотидных праймеров должны быть совместимы друг с другом, то есть не образовывать вторичных структур, чтобы избежать ингибирования 26 амплификации некоторых геномных фрагментов, а также амплификации дополнительных локусов при проведении мультиплексной ПЦР.

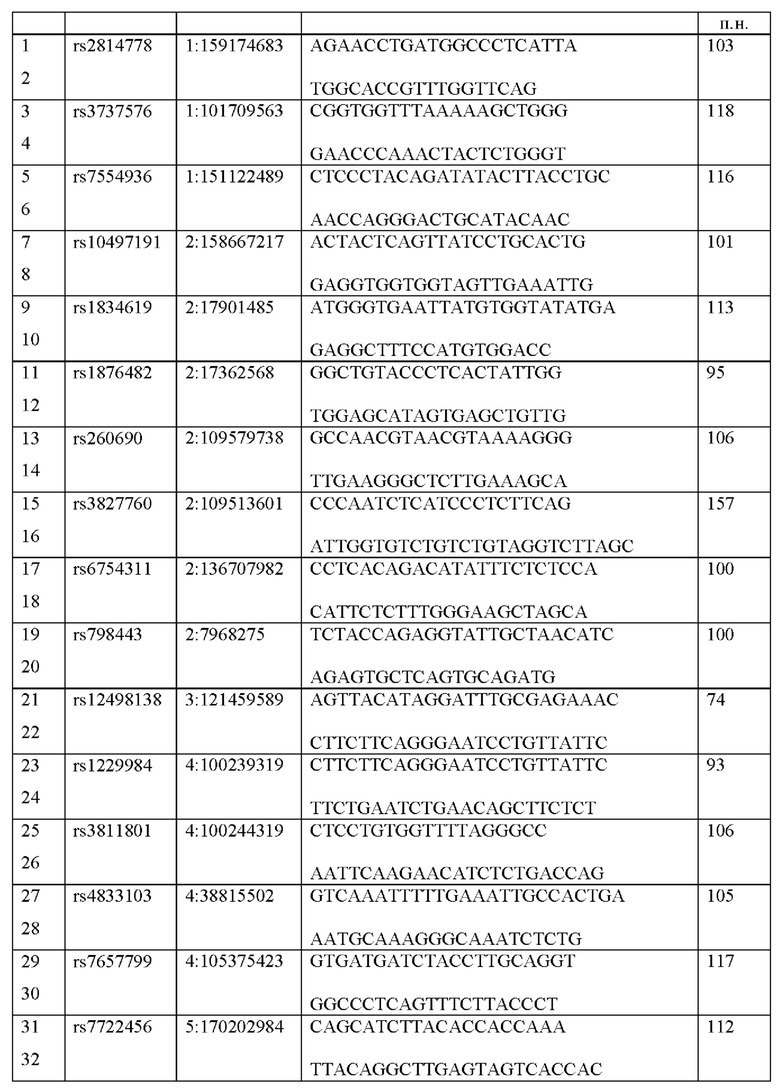
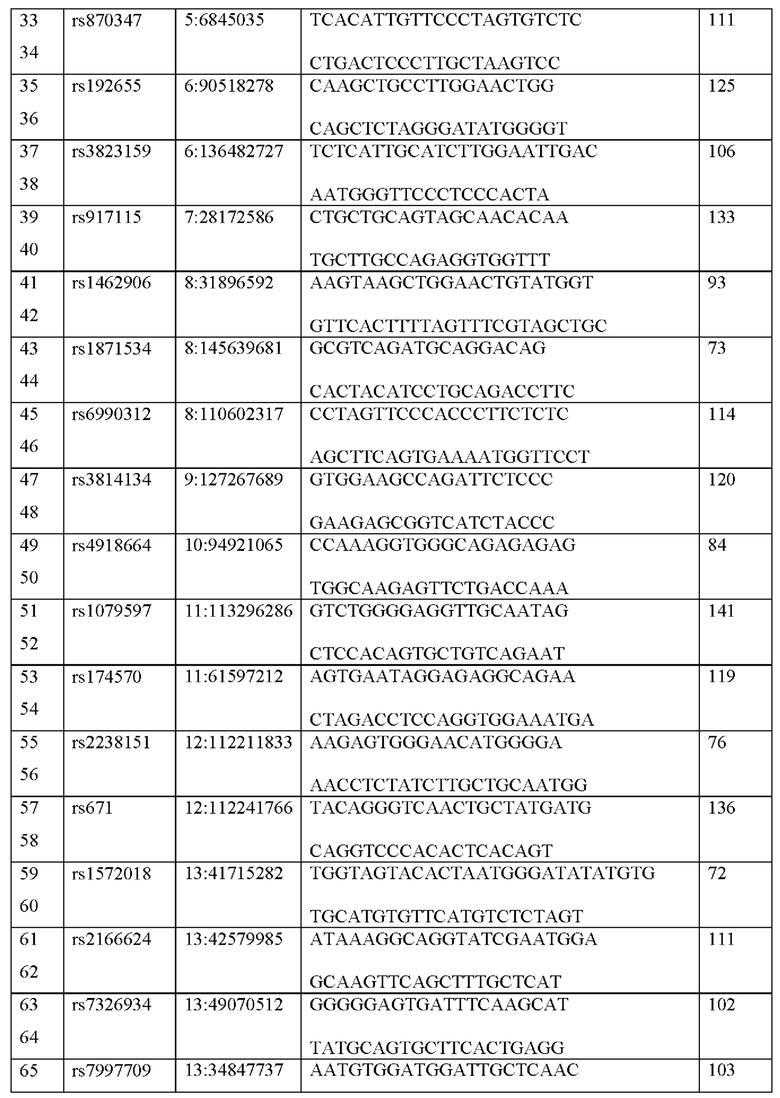
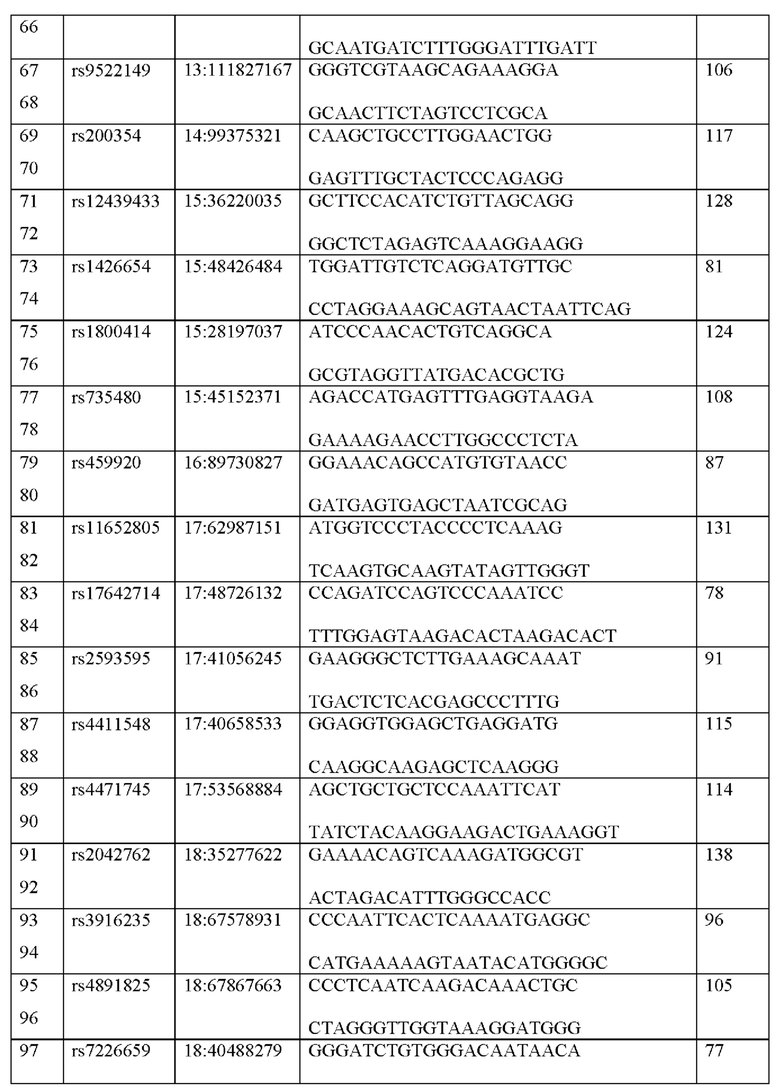
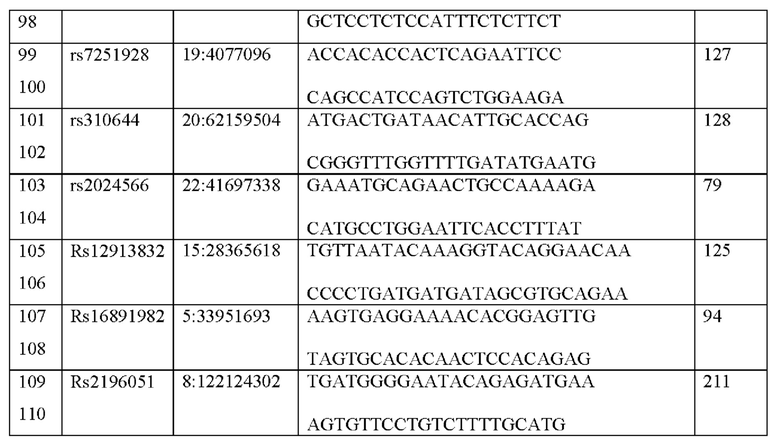
Авторами была проведена серия испытаний набора реактивов. Для этого было проведено полногеномное секвенирование образцов ДНК различных индивидов и было выявлено полное соответствие всех генотипов секвенированных образцов с генотипами всех 55 маркеров в ПЦР продуктах, полученными с помощью данного набора олигонуклеотидов на тех же образцах ДНК.
Возможность точного определения генотипов для всех 55 маркеров была показана для более чем 40 различных образцов ДНК, в том числе для образцов с сильно деградированной ДНК и на малых количествах ДНК. Таким образов, авторами для выбранных молекулярных маркеров была разработана панель из 55 пар оригинальных олигонуклеотидных праймеров, которые позволяют амплифицировать целевые фрагменты генома человека с выбранными маркерами для работы с образцами деградированной ДНК и малых количеств ДНК.
Осуществление способа
Пример генотипирования биологического образца.
Возможность определения вероятного этногеографического происхождения человека показана на образцах полногеномной ДНК и деградированной геномной ДНК индивидов известного различного этнического происхождения путем проведения мультиплексного ПЦР-анализа с последующим масштабным параллельным секвенированием полученных ДНК-образцов.
Для проведения ДНК-идентификации используется образец ДНК в количестве не менее 100 пкг, выделенная из любого биоматериала человека.
Получение ДНК.
ДНК может быть получена из любой ткани человека, содержащей клеточные ядра, например, лейкоцитов, волосяных фолликулов, клеток буккального эпителия и др. Пригодная для проведения ПЦР ДНК может быть выделена из образа при помощи любого ранее широко применяемого метода выделения ДНК должен быть в равной степени эффективным. В частности, есть несколько готовых наборов для выделения ДНК из различных тканей человека (например, представленных фирмой Qiagen)
Принцип определения вероятного этногеографического происхождения неизвестного индивида.
Определение вероятного этногеографического происхождения индивида включает в себя четыре этапа:
- Мультиплексная ПЦР геномной ДНК с использованием смеси специфических олигонуклеотидных праймеров;
- Приготовление фрагментных библиотек из полученных ПЦР-продуктов для секвенирования на платформе Illumina;
- Проведение широкомасштабного параллельного секвенирования полученных библиотек;
- Анализ данных секвенирования и интерпретация результатов.
Амплификация участков генома, содержащих исследуемые маркеры.
Амплификация участков генома, содержащих исследуемые маркеры, может быть осуществлена с помощью наборов Multiplex PCR kit компании «Qiagen», на приборах GeneAmp PCR System 9700 Thermal Cycler компании «Applied Biosystems».
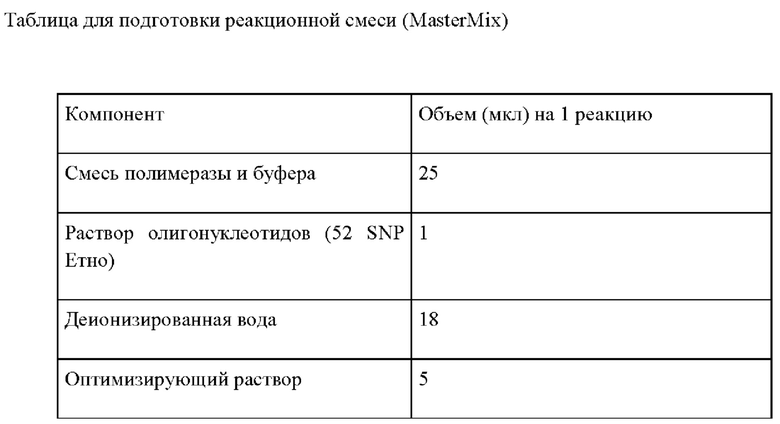
Состав реакционной смеси для мультиплексной ПЦР:
- Раствор олигонуклеотидов для мультиплексной ПЦР (в конечной концентрации от 1 нМ до 0,5 мкМ
- Смесь полимеразы и буфера
- Деионизированная вода
- Оптимизирующий раствор
- исследуемая ДНК
Набор олигонуклеотидных праймеров адаптирован для использования ДНК в количестве 1 нг, оптимальное рекомендуемое количество анализируемой ДНК от 100 пг до 10 нг. При необходимости, образец ДНК следует развести в буфере ЕВ.
Также необходимо проводить параллельно контрольную реакцию (отрицательный контроль), где вместо ДНК добавляется деионизированная вода.
Амплификацию следует проводить в пробирках, стрипах, или планшетах предназначенных для ПЦР.
В отдельной стерильной пробирке смешать из расчета на одну реакцию все необходимые компоненты, согласно расчетной таблице (пробирка с раствором MasterMix).
После приготовления, смесь для амплификации помещают в амплификатор и выставляется программа:
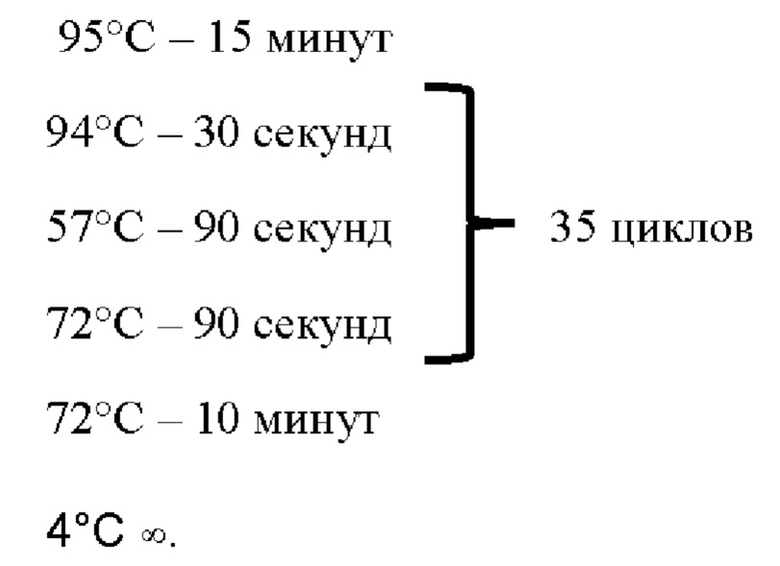
Наличие продуктов амплификации исследуемой ДНК и отсутствие продуктов амплификации в отрицательном контроле проверяют с помощью электрофореза в агарозном геле. Продукты амплификации смесь очищают от остаточных праймеров с помощью соответствующих мини-колонок (например, Qiagen MinElute system) в соответствии с инструкцией к используемому набору реагентов. Полученные в результате мультиплексной амплификации очищенные ПЦР продукты, содержащие маркеры для определения вероятного этногеографического происхождения человека используются в качестве матрицы для приготовления фрагментных геномных библиотек для секвенирования.
Приготовление фрагментных библиотек.
Предварительно необходимо провести измерение концентрации очищенных ПЦР-фрагментов, полученных на предыдущем этапе мультиплексной амплификации на приборах NanoDrop или Qubit (ThermoFisher) в соответствии с инструкцией к прибору. Для приготовления фрагментных библиотек оптимально использовать около 300 нг ДНК очищенных ПЦР-продуктов мультиплексной амплификации. Приготовление фрагментных геномных библиотек проводят с использованием наборов реагентов Illumina®. TruSeq® DNA PCR-Free library prep kit в соответствии с инструкцией к набору. Для секвенирования нескольких образцов фрагментных библиотек одновременно необходимо использовать индексированные адаптеры (например, Dual index adaptors Illumina). Для каждого образца библиотеки используется отдельный индексируемый адаптер, который необходимо записать в таблицу для дальнейшего учета при анализе результатов секвенирования нескольких образцов фрагментных библиотек. Для приготовления фрагментных библиотек рекомендуется/возможно использовать 1/2 от объема всех реагентов, входящих в набор Illumina®. TruSeq® DNA PCR-Free library prep kit, прописанного в инструкции к набору.
Этапы 1-3 являются рекомендованными, но не обязательными.
1 Очистка фрагментных библиотек: необходимо нанести весь объем элюата в одну лунку 2% агарозного геля с красителем SYBR Gold (например, с использованием готового 2%-ного E-gel (Invitrogen)). Провести электрофорез с использованием маркера молекулярных весов 50 bp Ladder в течение 10 минут (в случае использования системы e-gel (Invitrigen)).
2 с использованием стерильного одноразового скальпеля вырезать кусочки геля, с фрагментами библиотеки от 220 п. н. и больше. Светящиеся фрагменты длиной меньше 150 п. н. соответствуют димерам адаптеров и не должны быть использованы для дальнейшего анализа.
3 Провести очистку вырезанных фрагментов ДНК с использованием набора реагентов MinElute gele extraction kit (Qiagen). Эллюировать в 20-22 мкл буфера ЕВ (Qiagen).
Приготовленные описанным выше способом фрагментные библиотеки можно напрямую использовать для секвенирования на технологической платформе Illumina, предварительно проведя оценку качества полученных фрагментных библиотек.
Качественную и количественную оценку фрагментных библиотек можно проводить стандартными методами, рекомендованными фирмой производителем платформ для секвенирования Illumina. Для оптимизации рабочего процесса рекомендуем использовать следующие этапы: измерить концентрации фрагментных библиотек на приборе Qubit с использованием набора реагентов HS. Для дальнейшего секвенирования рекомендуется использовать фрагментные библиотеки с концентрацией не менее 2 нМ/мкл.
Перед секвенированием необходимо провести мультиплексирование библиотек в эквимолярных количествах (объединение библиотек для нескольких образцов). Не допускается объединение в один пул библиотек с одинаковыми индексами.
Приготовленные фрагментные библиотеки секвенируют методом параллельного широкомасштабного секвенирования на технологической платформе Illumina в парноконцевом режиме с длинной прочтения не менее 75+75 п.о. Рекомендуемый прибор секвенатор MiSeq (Illumina).
Анализ результатов секвенирования.
Анализ результатов секвенирования проводят следующим образом:
- Картировать полученные прочтения на референсный геном человека (GRCh37) с помощью программы bwamem или аналогичной.
- Определить генотипы по исследуемым маркерам с помощью программ GATK, freebayes или аналогичных. Список маркеров и координаты (bed-файл см в Приложении 2).
- Загрузить файл в формате txt на сайт https://frog.med.yale.edu/FrogKB/FrogServlet, в разделе AISNP, KiddLab - Set of 55 AISNPs согласно представленной там инструкции.
Литературные источники.
1. Rosenberg N.A. et al. Genetic structure of human populations. // Science. 2002. Vol. 298, №5602. P. 2381-2385.
2. Hoggart C.J. et al. Control of confounding of genetic associations in stratified populations. // Am. J. Hum. Genet. 2003. Vol. 72, №6. P. 1492-1504.
3. Marchini J. et al. The effects of human population structure on large genetic association studies. //Nat. Genet. 2004. Vol. 36, №5. P. 512-517.
4. Jobling M.A., Gill P. Encoded evidence: DNA in forensic analysis. // Nat. Rev. Genet. 2004. Vol. 5, №10. P. 739-751.
5. Yang N. et al. Examination of ancestry and ethnic affiliation using highly informative diallelic DNA markers: application to diverse and admixed populations and implications for clinical epidemiology and forensic medicine. // Hum. Genet. 2005. Vol. 118, №3-4. P. 382 392.
6. King J.L. et al. High-quality and high-throughput massively parallel sequencing of the human mitochondrial genome using the Illumina MiSeq. // Forensic Sci. Int. Genet. 2014. Vol. 12. P. 128-135.
7. Jobling M.A., Tyler-Smith C. The human Y chromosome: an evolutionary marker comes of age. //Nat. Rev. Genet. 2003. Vol. 4, №8. P. 598-612.
8. Vigilant L. et al. African populations and the evolution of human mitochondrial DNA. // Science. 1991. Vol. 253, №5027. P. 1503-1507.
9. Jin L., Chakraborty R. Population structure, stepwise mutations, heterozygote deficiency and their implications in DNA forensics. // Heredity (Edinb). 1995. Vol. 74 (Pt 3). P. 274-285.
10. Smith M.W. et al. Markers for mapping by admixture linkage disequilibrium in African 96 American and Hispanic populations. // Am. J. Hum. Genet. 2001. Vol. 69, №5. P. 1080-1094.
11. Terwilliger J.D., Hiekkalinna T. An utter refutation of the "fundamental theorem of the НарМар". // Eur. J. Hum. Genet. 2006. Vol. 14, №4. P. 426-137.
12. Auton A. et al. A global reference for human genetic variation // Nature. 2015. Vol. 526, №7571. P. 68-74.
13 Kidd K.K. et al. Progress toward an efficient panel of SNPs for ancestry inference. // Forensic Sci. Int. Genet. 2014. Vol. 10. P. 23-32.
14 Phillips C. et al. Inferring ancestral origin using a single multiplex assay of ancestryinformative marker SNPs. // Forensic Sci. Int. Genet. 2007. Vol. 1, №3-4. P. 273-280.
15 Nievergelt CM. et al. Inference of human continental origin and admixture proportions using a highly discriminative ancestry informative 41-SNP panel. // Investig. Genet. 2013. Vol. 4, №1. P. 13.
16 Claes P. et al. Modeling 3D facial shape from DNA. // PLoS Genet. Public Library of Science, 2014. Vol. 10, №3. P. el004224.
17. Wei Y.-L. et al. A single-tube 27-plex SNP assay for estimating individual ancestry and admixture from three continents // Int. J. Legal Med. 2016. Vol. 130, №1. P. 27-37.
18 173. Sachidanandam R. et al. A map of human genome sequence variation containing 1.42 million single nucleotide polymorphisms. // Nature. 2001. Vol. 409, №6822. P. 928 933.
19 174. Thorisson G.A. The International НарМар Project Web site // Genome Res. 2005. Vol. 15, №11. P. 1592-1593.
20 Haider I. et al. A panel of ancestry informative markers for estimating individual biogeographical ancestry and admixture from four continents: utility and applications. // Hum. Mutat. 2008. Vol. 29, №5. P. 648-658.
21 Rosenberg N.A. et al. Informativeness of genetic markers for inference of ancestry. // Am. J. Hum. Genet. 2003. Vol. 73, №6. P. 1402-1422.
22 Chen H.-D. et al. Divergence and Shannon information in genomes. // Phys. Rev. Lett. 2005. Vol. 94, №17. P. 178103.
23 WRIGHT S. Genetical structure of populations. // Nature. 1950. Vol. 166, №4215. P. 247-249.
24 Ding L. et al. Comparison of measures of marker infbrmativeness for ancestry and admixture mapping. // BMC Genomics. 2011. Vol. 12. P. 622.
25 Kosoy R. et al. Ancestry informative marker sets for determining continental origin and admixture proportions in common populations in America. // Hum. Mutat. 2009. Vol. 30, №1. P. 69-78.
26 Kersbergen P. et al. Developing a set of ancestry-sensitive DNA markers reflecting continental origins of humans // BMC Genet. 2009. Vol. 10, №1. P. 69.
27 Fondevila M. et al. Revision of the SNPforID 34-plex forensic ancestry test: Assay enhancements, standard reference sample genotypes and extended population studies // Forensic Sci. Int. Genet. 2013. Vol. 7, №1. P. 63-74.
28 Gettings K.B. et al. A 50-SNP assay for biogeographic ancestry and phenotype prediction in the U.S. population. //Forensic Sci. Int. Genet. 2014. Vol. 8, №1. P. 101-108.
29 Elhaik, E., Tatarinova, Т., Chebotarev, D. et al. Geographic population structure analysis of worldwide human populations infers their biogeographical origins. Nat Commun 5, 3513 (2014). https://doi.org/10.1038/ncomms4513
30. Wollstein A, Lao O. Detecting individual ancestry in the human genome. Investig Genet. 2015 May 1;6:7. doi: 10.1186/s13323-015-0019-x.
31. Hall JB, Bush WS. Analysis of Heritability Using Genome-Wide Data. Curr Protoc Hum Genet. 2016 Oct 11; 91:1.30.1-1.30.10. doi: 10.1002/cphg.25. PMID: 27727439; PMCID: PMC5127448.
32. Wong, Emily H. M. et al. "Reconstructing genetic history of Siberian and Northeastern European populations." Genome research 27 1 (2017): 1-14
33. Khrunin AV, Khokhrin DV, Filippova IN, Esko T, Nelis M, Bebyakova NA, et al. (2013) A Genome-Wide Analysis of Populations from European Russia Reveals a New Pole of Genetic Diversity in Northern Europe. PLoS ONE 8(3): e58552. https://doi.org/10.1371/iournal.pone.0058552
Перечень нуклеотидных последовательностей
SEQ ID
SEQ ID NO:1
AGAACCTGATGGCCCTCATTA
SEQ ID NO:2
TGGCACCGTTTGGTTCAG
SEQ ID NO:3
CGGTGGTTTAAAAAGCTGGG
SEQ ID NO:4
GAACCCAAACTACTCTGGGT
SEQ ID NO:5
CTCCCTACAGATATACTTACCTGC
SEQ ID NO:6
AACCAGGGACTGCATACAAC
SEQ ID NO:7
ACTACTCAGTTATCCTGCACTG
SEQ ID NO:8
GAGGTGGTGGTAGTTGAAATTG
SEQ ID NO:9
ATGGGTGAATTATGTGGTATATGA
SEQ ID NO:10
GAGGCTTTCCATGTGGACC
SEQ ID NO:11
GGCTGTACCCTCACTATTGG
SEQ ID NO:12
TGGAGCATAGTGAGCTGTTG
SEQ ID NO:13
GCCAACGTAACGTAAAAGGG
SEQ ID NO:14
TTGAAGGGCTCTTGAAAGCA
SEQ ID NO:15
CCCAATCTCATCCCTCTTCAG
SEQ ID NO:16
ATTGGTGTCTGTCTGTAGGTCTTAGC
SEQ ID NO:17
CCTCACAGACATATTTCTCTCCA
SEQ ID NO:18
CATTCTCTTTGGGAAGCTAGCA
SEQ ID NO:19
TCTACCAGAGGTATTGCTAACATC
SEQ ID NO:20
AGAGTGCTCAGTGCAGATG
SEQ ID NO:21
AGTTACATAGGATTTGCGAGAAAC
SEQ ID NO:22
CTTCTTCAGGGAATCCTGTTATTC
SEQ ID NO:23
CTTCTTCAGGGAATCCTGTTATTC
SEQ ID NO:24
TTCTGAATCTGAACAGCTTCTCT
SEQ ID NO:25
CTCCTGTGGTTTTAGGGCC
SEQ ID NO:26
AATTCAAGAACATCTCTGACCAG
SEQ ID NO:27
GTCAAATTTTTGAAATTGCCACTGA
SEQ ID NO:28
AATGCAAAGGGCAAATCTCTG
SEQ ID NO:29
GTGATGATCTACCTTGCAGGT
SEQ ID NO:30
GGCCCTCAGTTTCTTACCCT
SEQ ID NO:31
CAGCATCTTACACCACCAAA
SEQ ID NO:32
TTACAGGCTTGAGTAGTCACCAC
SEQ ID NO:33
TCACATTGTTCCCTAGTGTCTC
SEQ ID NO:34
CTGACTCCCTTGCTAAGTCC
SEQ ID NO:35
CAAGCTGCCTTGGAACTGG
SEQ ID NO:36
CAGCTCTAGGGATATGGGGT
SEQ ID NO:37
TCTCATTGCATCTTGGAATTGAC
SEQ ID NO:38
AATGGGTTCCCTCCCACTA
SEQ ID NO:39
CTGCTGCAGTAGCAACACAA
SEQ ID NO:40
TGCTTGCCAGAGGTGGTTT
SEQ ID NO:41
AAGTAAGCTGGAACTGTATGGT
SEQ ID NO:42
GTTCACTTTTAGTTTCGTAGCTGC
SEQ ID NO:43
GCGTCAGATGCAGGACAG
SEQ ID NO:44
CACTACATCCTGCAGACCTTC
SEQ ID NO:45
CCTAGTTCCCACCCTTCTCTC
SEQ ID NO:46
AGCTTCAGTGAAAATGGTTCCT
SEQ ID NO:47
GTGGAAGCCAGATTCTCCC
SEQ ID NO:48
GAAGAGCGGTCATCTACCC
SEQ ID NO:49
CCAAAGGTGGGCAGAGAGAG
SEQ ID NO:50
TGGCAAGAGTTCTGACCAAA
SEQ ID NO:51
GTCTGGGGAGGTTGCAATAG
SEQ ID NO:52
CTCCACAGTGCTGTCAGAAT
SEQ ID NO:53
AGTGAATAGGAGAGGCAGAA
SEQ ID NO:54
CTAGACCTCCAGGTGGAAATGA
SEQ ID NO:55
AAGAGTGGGAACATGGGGA
SEQ ID NO:56
AACCTCTATCTTGCTGCAATGG
SEQ ID NO:57
TACAGGGTCAACTGCTATGATG
SEQ ID NO:58
CAGGTCCCACACTCACAGT
SEQ ID NO:59
TGGTAGTACACTAATGGGATATATGTG
SEQ ID NO:60
TGCATGTGTTCATGTCTCTAGT
SEQ ID NO:61
ATAAAGGCAGGTATCGAATGGA
SEQ ID NO:62
GCAAGTTCAGCTTTGCTCAT
SEQ ID NO:63
GGGGGAGTGATTTCAAGCAT
SEQ ID NO:64
TATGCAGTGCTTCACTGAGG
SEQ ID NO:65
AATGTGGATGGATTGCTCAAC
SEQ ID NO:66
GCAATGATCTTTGGGATTTGATT
SEQ ID NO:67
GGGTCGTAAGCAGAAAGGA
SEQ ID NO:68
GCAACTTCTAGTCCTCGCA
SEQ ID NO:69
CAAGCTGCCTTGGAACTGG
SEQ ID NO:70
GAGTTTGCTACTCCCAGAGG
SEQ ID NO:71
GCTTCCACATCTGTTAGCAGG
SEQ ID NO:72
GGCTCTAGAGTCAAAGGAAGG
SEQ ID NO:73
TGGATTGTCTCAGGATGTTGC
SEQ ID NO:74
CCTAGGAAAGCAGTAACTAATTCAG
SEQ ID NO:75
ATCCCAACACTGTCAGGCA
SEQ ID NO:76
GCGTAGGTTATGACACGCTG
SEQ ID NO:77
AGACCATGAGTTTGAGGTAAGA
SEQ ID NO:78
GAAAAGAACCTTGGCCCTCTA
SEQ ID NO:79
GGAAACAGCCATGTGTAACC
SEQ ID NO:80
GATGAGTGAGCTAATCGCAG
SEQ ID NO:81
ATGGTCCCTACCCCTCAAAG
SEQ ID NO:82
TCAAGTGCAAGTATAGTTGGGT
SEQ ID NO:83
CCAGATCCAGTCCCAAATCC
SEQ ID NO:84
TTTGGAGTAAGACACTAAGACACT
SEQ ID NO:85
GAAGGGCTCTTGAAAGCAAAT
SEQ ID NO:86
TGACTCTCACGAGCCCTTTG
SEQ ID NO:87
GGAGGTGGAGCTGAGGATG
SEQ ID NO:88
CAAGGCAAGAGCTCAAGGG
SEQ ID NO:89
AGCTGCTGCTCCAAATTCAT
SEQ ID NO:90
TATCTACAAGGAAGACTGAAAGGT
SEQ ID NO:91
GAAAACAGTCAAAGATGGCGT
SEQ ID NO:92
ACTAGACATTTGGGCCACC
SEQ ID NO:93
CCCAATTCACTCAAAATGAGGC
SEQ ID NO:94
CATGAAAAAGTAATACATGGGGC
SEQ ID NO:95
CCCTCAATCAAGACAAACTGC
SEQ ID NO:96
CTAGGGTTGGTAAAGGATGGG
SEQ ID NO:97
GGGATCTGTGGGACAATAACA
SEQ ID NO:98
GCTCCTCTCCATTTCTCTTCT
SEQ ID NO:99
ACCACACCACTCAGAATTCC
SEQ ID NO:100
CAGCCATCCAGTCTGGAAGA
SEQ ID NO:101
ATGACTGATAACATTGCACCAG
SEQ ID NO:102
CGGGTTTGGTTTTGATATGAATG
SEQ ID NO:103
GAAATGCAGAACTGCCAAAAGA
SEQ ID NO:104
CATGCCTGGAATTCACCTTTAT
SEQ ID NO:105
TGTTAATACAAAGGTACAGGAACAA
SEQ ID NO:106
CCCCTGATGATGATAGCGTGCAGAA
SEQ ID NO:107
AAGTGAGGAAAACACGGAGTTG
SEQ ID NO:108
TAGTGCACACAACTCCACAGAG
SEQ ID NO:109
TGATGGGGAATACAGAGATGAA
SEQ ID NO:110
AGTGTTCCTGTCTTTTGCATG
Изобретение относится к области биотехнологии, судебной медицины и криминалистики, генетическим исследованиям в области идентификации человека. Представлен способ получения молекулярных маркеров для определения вероятного этногеографического происхождения человека и набор синтетических олигонуклеотидных праймеров для проведения мультиплексной полимеразной цепной реакции (ПЦР). Набор для генотипирования включает композицию, образованную 55 парами синтетических оригинальных олигонуклеотидных праймеров для локусов в геноме человека, включающих SNP-маркеры. Синтетические олигонуклеотидные праймеры подобраны таким образцом, чтобы ПЦР-продукт имел минимальную длину, меньше 150 п. н. Изобретение относится также к способу генотипирования молекулярных маркеров для определения вероятного этногеографического происхождения человека методом мультиплексной амплификации для работы с образцами деградированной ДНК. Предложено применение способа получения молекулярных маркеров для определения вероятного этногеографического происхождения человека в российской популяции. Изобретение позволяет определять вероятное этногеографическое происхождение человека при работе с образцами деградированной ДНК с высокой чувствительностью, специфичностью и точностью. 4 н. и 6 з.п. ф-лы, 2 табл., 1 пр.
1. Способ получения молекулярных маркеров для определения вероятного этногеографического происхождения человека методом мультиплексной амплификации для работы с образцами деградированной ДНК, характеризующийся тем, что для генотипирования используется композиция из 55 пар оригинальных синтетических олигонуклеотидных праймеров для проведения мультиплексной полимеразной цепной реакции (ПЦР), выбранных из группы SEQ ID NO: 1 - SEQ ID NO: 110, фланкирующих участки ДНК, содержащие однонуклеотидные вариации (SNP), ПЦР-продукты, соответствующие выбранным праймерам, представляют собой маркеры.
2. Способ определения вероятного этногеографического происхождения человека, включающий генетическое исследование биологического образца, содержащего ДНК, с использованием мультиплексной полимеразно-цепной реакции (мультиплексной ПЦР) и последующее определение нуклеотидной последовательности продуктов амплификации, содержащих SNP-маркеры определения вероятного этногеографического происхождения, полученные способом по п. 1.
3. Способ определения вероятного этногеографического происхождения человека по п. 2, отличающийся тем, что олигонуклеотидные праймеры для мультиплексной ПЦР выбраны из композиции SEQ ID NO: 1 - SEQ ID NO: 110.
4. Способ определения вероятного этногеографического происхождения человека по п. 2, отличающийся тем, что размер полученных ПЦР-продуктов составляет не более 150 п. н.
5. Способ определения вероятного этногеографического происхождения человека по п. 2, отличающийся тем, что полученные в результате мультиплексной амплификации ПЦР-продукты, содержащие молекулярные SNP-маркеры определения вероятного этногеографического происхождения, используются в качестве матрицы для различных методов генетического определения нуклеотидной последовательности.
6. Способ определения вероятного этногеографического происхождения человека по п. 2, отличающийся тем, что полученные в результате мультиплексной амплификации ПЦР-продукты, содержащие молекулярные SNP-маркеры определения вероятного этногеографического происхождения, используются в качестве матрицы для приготовления геномных библиотек, предназначенных для определения нуклеотидной последовательности секвенированием по методу "массового параллельного секвенирования" (NGS).
7. Способ определения вероятного этногеографического происхождения человека по п. 2, отличающийся тем, что определение нуклеотидной последовательности продуктов амплификации (ПЦР-продуктов) проводят секвенированием по методу "массового параллельного секвенирования" (NGS), секвенированием по методу Сэнгера, методом TaqMan Real-Time PCR, а также проводят любым доступным способом определения нуклеотидной последовательности.
8. Способ определения вероятного этногеографического происхождения человека по п. 2, отличающийся тем, что исследуемый биологический образец содержит деградированную ДНК.
9. Набор олигонуклеотдов для генотипирования индивида способом по п. 1, используемый для детектирования присутствия молекулярных SNP-маркеров, определяющий вероятное этногеографическое происхождение человека, включающий синтетические оригинальные олигонуклеотиды с нуклеотидными последовательностями SEQ ID NO: 1 - SEQ ID NO: 110.
10. Применение способа по п. 1 для определения вероятного этногеографического происхождения индивида, происходящего из российских популяций.
| KYRUNIN A.V | |||
| et al | |||
| Genome-Wide analysis of populations from European Russia Reveals a New Pole of Genetic Diversity in Northem Europe, Article in PloS ONE, March 2013, v/8, p.1-9, file://C:/Users/otd1333/Downloads/Genome-Wide Analysis of populations from Europea.pdf | |||
| KIDD K., K | |||
| et al | |||
| Progress toward an efficient panel of SNPs for ancertry |
Авторы
Даты
2023-07-18—Публикация
2021-11-10—Подача