Область техники, к которой относится изобретение
Изобретение относится к молекулярной биологии, микробиологии и медицине и обеспечивает внутривидовое и межвидовое генетическое типирование микобактерий туберкулезного комплекса.
Уровень техники
Туберкулез (ТБ) остается одним из наиболее опасных инфекционных заболеваний, уносящих миллионы жизней в год (Floyd, K., Glaziou, P., Zumla, A., Raviglione, М. (2018) The global tuberculosis epidemic and progress in care, prevention, and research: an overview in year 3 of the End ТВ era. Lancet Respir Med, 6, 299-314). Из-за быстрого трансконтинентального распространения инфекции и особенно частой коинфекции с ВИЧ (Adhikari, N., Bhattarai, R.B., Basnet, R., Joshi, L.R., Tinkari, B.S., Thapa, A., Joshi, B. (2022) Prevalence and associated risk factors for tuberculosis among people living with HIV in Nepal. PloS one, 17, e0262720; Ashenafi, S., Rao Muwa, J., Mily, A., Snail, J., Zewdie, M., Chanyalew, M, Rehn, A., Rahman, S., Aseffa, G., Bekele, A., Aderaye, G., Lema, В., Svensson, M., Brighenti, S. (2022) Immunosuppressive features of the microenvironment in lymph nodes granulomas from tuberculosis and HIV co-infected patients. Am J Path S0002-9440(22)00012-8) число новых случаев имеет тенденцию к увеличению как в промышленно развитых странах, так и в менее развитых странах (Daley, C.L. (2019) The Global Fight Against Tuberculosis. Thorac Surg Clin, 29, 19-25).
Помимо своевременной диагностики, ключевыми факторами в борьбе с туберкулезом являются быстрое выявление возбудителя, тестирование на лекарственную чувствительность, немедленное ограничение передачи и адекватное медикаментозное лечение. Эпидемиологический анализ, основанный на молекулярном генотипировании патогена, является мощным инструментом в идентификации источников инфекции и путей передачи, которые позволяют принимать соответствующие меры (Driscoll, J.R. (2009) Spoligotyping for molecular epidemiology of the Mycobacterium tuberculosis complex. Meth Mol Biol (Clifton, N.J.), 551, 117-128; Kamerbeek, J., Schouls, L., Kolk, A., Van Agterveld, M., Van Soolingen, D., Kuijper, S., Bunschoten, A., Molhuizen, H., Shaw, R., Goyal, M, Van Embden, J. (1997) Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology. J Clin Microbiol, 35, 907-914; Tejashree, A., Mahesh, P.A., Krishna Karthik, M., Nirmala Azeem, A., Reddy, R., Ravichandra, C, Nagaraja, S.B. (2022) Era of ТВ elimination: Growing need to understand diversities of Mycobacterium tuberculosis lineages!. Indian J Tuberc, 69, 79-84).
Однако, если существующие экспресс-методики определения антибиотикоустойчивости, основанные на ПЦР, связаны с анализом определенной генной мишени (при известном молекулярно-генетическом механизме устойчивости), то ситуация с внутривидовым типированием не столь однозначна в силу отсутствия универсального метода, хорошо применимого для большинства туберкулезных штаммов. Принимая во внимание региональные особенности эндемичных штаммов, принадлежащих к различным эволюционным типам, что отражается на их молекулярно-биологических свойствах, часто приходится выбирать оптимальный подход типирования для каждого географического региона (Diriba, В., Berkessa, Т., Mamo, G., Tedla, Y., Ameni, G. (2013) Spoligotyping of multidrug-resistant Mycobacterium tuberculosis isolates in Ethiopia. Int J Tuberc Lung Dis, 17, 246-250; Haile, В., Tafess, K., Zewude, A., Yenew, В., Siu, G., Ameni, G. (2019) Spoligotyping and drug sensitivity of Mycobacterium tuberculosis isolated from pulmonary tuberculosis patients in the Arsi Zone of southeastern Ethiopia. New Microbes New Infect, 33,100620; Ramazanzadeh, R., Shakib, P., Rouhi, S., Mohammadi, В., Mohajeri, P., Borji, S. (2020) Molecular epidemiology of Mycobacterium tuberculosis isolates in Iran using spoligotyping. New Microbes New Infect, 38, 100767).
Сравнению возможностей различных методов типирования посвящено довольно много работ (Ei, P.W., Aung, W.W., Lee, J.S., Choi, G.E., Chang, C.L. (2016) Molecular Strain Typing of Mycobacterium tuberculosis: a Review of Frequently Used Methods. J Korean Med Sci, 31,1673-1683; Masoud, K., Araj, G.F., Reslan, L., Fadlallah, S., Wehbe, M., Itani, L., Avedissian, A., Dbaibo, G., Saade, A., Refregier, G., Sola, C, Matar, G.M. (2020) Spoligotyping of Mycobacterium tuberculosis isolates using Luminex®-based method in Lebanon. J Infect Dev Ctries, 14, 878-885; Ravansalar, H., Tadayon, K., Ghazvini, K. (2016) Molecular typing methods used in studies of Mycobacterium tuberculosis in Iran: a systematic review. Iran J Microbiol, 8, 338-346). Кроме того, предложены комбинации нескольких методов, которые, по мнению авторов, позволяют повысить уровень дифференцировки и осуществлять более качественный эпидемиологический мониторинг (Parreiras, P.M., Andrade, G.I., Nascimento, Т., Oelemann, M.C., Gomes, H.M., Alencar, A.P., Assis, R.A., Mota, P.M., Pereira, M.A., Lobato, F.C., Lage, A.P., Suffys, P.N. (2012) Spoligotyping and variable number tandem repeat analysis of Mycobacterium bovis isolates from cattle in Brazil. Mem Inst Oswaldo Cruz, 107, 64-73; Shi, J., Zheng, D., Zhu, Y., Ma, X., Wang, S., Li, H., Xing, J. (2018) Role of MIRU-VNTR and spoligotyping in assessing the genetic diversity of Mycobacterium tuberculosis in Henan Province, China. BMC Infect Dis, 18, 447). В настоящее время широко распространенными являются методы, основанные на фингерпринтинге различных геномных мишеней, таких как IS 6110 (Dekhil, N., Skhairia, M.A., Mhenni, В., Ben Fradj, S., Warren, R., Mardassi, H. (2018) Automated IS6110-based fingerprinting of Mycobacterium tuberculosis: Reaching unprecedented discriminatory power and versatility. PloS One, 13, e0197913), PGRS (Yang, Z.H., Ijaz, K, Bates, J.H., Eisenach, K.D., Cave, M.D. (2000) Spoligotyping and polymorphic GC-rich repetitive sequence fingerprinting of Mycobacterium tuberculosis strains having few copies of IS6110. J Clin Microbiol, 38, 3572-3576), DR (Driscoll, J.R. (2009) Spoligotyping for molecular epidemiology of the Mycobacterium tuberculosis complex. Meth Mol Biol (Clifton, N.J.), 551, 117-128; Tejashree, A., Mahesh, P.A., Krishna Karthik, M., Nirmala Azeem, A., Reddy, R., Ravichandra, C, Nagaraja, S.B. (2022) Era of ТВ elimination: Growing need to understand diversities of Mycobacterium tuberculosis lineages!. Indian J Tuberc, 69, 79-84), 16S rDNA (Cheung, M.K., Lam, W.Y., Fung, W.Y., Law, P.T., Au, C.H., Nong, W., Kam, K.M., Kwan, H.S., Tsui, S.K. (2013) Sputum microbiota in tuberculosis as revealed by 16S rRNA pyrosequencing. PloS One, 8, e54574) и другие подходы; однако, основным их недостатком является необходимость использования сложного оборудования для анализа получаемых электрофорезных профилей, а также необходимость в предварительном подращивании культуры (Merker, М., Kohl, Т.А., Niemann, S., Supply, P. (2017) The Evolution of Strain Typing in the Mycobacterium tuberculosis Complex. Adv Exp Med Biol, 1019, 43-78).
Это одна из причин, обусловивших распространение новых, основанных на ПЦР, методов типирования. Одним из таких методов является т.н. сполиготипирование (от. англ. spacer oligonucleotide typing). Метод основан на анализе локуса прямых повторов микобактериального генома, описанного в 1991 г. Hermans и соавторами (Hermans, P.W.M., van Soolingen, D., Bik, E.M., de Haas, P.E.W., Dale, J.W., van Embden, J.D.A. (1991) The insertion element IS987 from Mycobacterium bovis BCG is located in a hot spot integration region for insertion elements in Mycobacterium tuberculosis complex strains. Infect Immun, 59, 2695-2705). Данный локус характеризуется наличием многократно повторяющихся участков длиной 36 н.п.идентичной последовательности, между которыми расположены уникальные спейсерные последовательности длиной от 24 до 41 н.п. Различные штаммы характеризуются наличием разного числа таких спейсерных последовательностей, что позволяет делать заключения о степени их родства и, соответственно, проводить эпидемиологические исследования. Изначально предложенный для внутривидового типирования М. tuberculosis (Groenen, Р.М.А., Bunschoten, А.Е., van Soolingen, D., van Embden, J.D.A. (1993) Nature of DNA polymorphism in the direct repeat cluster of Mycobacterium tuberculosis; application of strain differentiation by a novel typing method. Mol Microbiol, 10, 1057-1065), метод, основанный на гибридизации ПЦР-продуктов со специфичными к спейсерным участкам зондами, затем успешно распространен и на бовисные штаммы (Aranaz, A., Liebana, Е., Mateos, A., Dominguez, L., Vidal, D., Domingo, M., Gonzolez, О., Rodriguez-Ferri, E.F., Bunschoten, A.E., Van Embden, J.D., Cousins, D. (1996) Spacer oligonucleotide typing of Mycobacterium bovis strains from cattle and other animals: a tool for studying epidemiology of tuberculosis. J Clin Microbiol, 34, 2734-2740; Cousins, D., Williams, S., Liebana, E., Aranaz, A., Bunschoten, A., Van Embden, J., Ellis, T. (1998) Evaluation of four DNA typing techniques in epidemiological investigations of bovine tuberculosis. J Clin Microbiol, 36, 168-178). К настоящему времени предпринимаются не только попытки стандартизации методики сполиготипирования (Dale, J.W., Brittain, D., Cataldi, A.A., Cousins, D., Crawford, J.T., Driscoll, J., Heersma, H., Lillebaek, Т., Quitugua, Т., Rastogi, N., Skuce, R.A., Sola, C, Van Soolingen, D., Vincent, V. (2001) Spacer oligonucleotide typing of bacteria of the Mycobacterium tuberculosis complex: recommendations for standardized nomenclature. Int J Tub Lung Dis, 5, 216-219), но и составлены обширные базы данных (Demay, С, Liens, В., Burguiere, Т., Hill, V., Couvin, D., Millet, J., Mokrousov, I., Sola, C, Zozio, Т., Rastogi, N. (2012) SITVITWEB - a publicly available international multimarker database for studying Mycobacterium tuberculosis genetic diversity and molecular epidemiology. Infect Genet Evol, 12, 755-766; Filliol, I., Driscoll, J.R., van Soolingen, D., et al. (2003) Snapshot of moving and expanding clones of Mycobacterium tuberculosis and their global distribution assessed by spoligotyping in an International Study. J Clin Microbiol, 41, 1963-1970). Стандартизированные базы данных содержат информацию о сорока трех (43) спейсерных последовательностях. Несмотря на отмечаемую многими авторами в ряде случаев меньшую дискриминирующую способность по сравнению с другими общепринятыми методами типирования (Bauer, J., Andersen, А.В., Kremer, K., Miorner, H. (1999) Usefulness of spoligotyping to discriminate IS6110 low-copy-number Mycobacterium tuberculosis complex strains cultured in Denmark. J Clin Microbiol, 37, 2602-2606; Kamerbeek, J., Schouls, L., Kolk, A., Van Agterveld, M., Van Soolingen, D., Kuijper, S., Bunschoten, A., Molhuizen, H., Shaw, R., Goyal, M., Van Embden, J. (1997) Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology. J Clin Microbiol, 35, 907-914; Kremer, K., Van Soolingen, D., Frothingham, R., Haas, W.H., Hermans, P.W.M., Martin, C, Palittapongarnpim, P., Plikaytis, B.B., Riley, L.W., Yakrus, M.A., Musser, J.M., Van Embden, J.D.A. (1999) Comparison of methods based on different molecular epidemiological markers for typing of Mycobacterium tuberculosis complex strains: interlaboratory study of discriminatory power and reproducibility. J Clin Microbiol, 37, 2607-2618; Wilson, S.M., Goss, S., Drobnievski, F. (1998) Evaluation of strategies for molecular fingerprinting for use in the routine work of a mycobacterium reference unit. J Clin Microbiol, 36, 3385-3388), отмечена дешевизна метода, простота его выполнения и интерпретации получаемых гибридизационных профилей (Cousins, D., Williams, S., Liebana, E., Aranaz, A., Bunschoten, A., Van Embden, J., Ellis, T. (1998) Evaluation of four DNA typing techniques in epidemiological investigations of bovine tuberculosis. J Clin Microbiol, 36, 168-178; Kremer, K., Van Soolingen, D., Frothingham, R., Haas, W.H., Hermans, P.W.M., Martin, C, Palittapongarnpim, P., Plikaytis, B.B., Riley, L.W., Yakrus, M.A., Musser, J.M., Van Embden, J.D.A. (1999) Comparison of methods based on different molecular epidemiological markers for typing of Mycobacterium tuberculosis complex strains: interlaboratory study of discriminatory power and reproducibility. J Clin Microbiol, 37, 2607-2618; Sola, C, Filliol, I., Legrand, E., Rastogi, N. (2000) Recent developments of spoligotyping as applied to the study of epidemiology, biodiversity and molecular phylogeny of the Mycobacterium tuberculosis complex. Pathol Biol (Paris), 48, 921-932). Это позволяет использовать сполиготипирование для первичной эпидемиологической оценки, затем, при необходимости, дополняя его методами с большей разрешающей способностью (Almeida, S.M., Malaspina, А.С., Leite, С, Saad, М. (2019) Usefulness of 3'- 5' IS6110-RFLP genotyping and spoligotyping of Mycobacterium tuberculosis isolated in a tertiary hospital: a retrospective study detecting unsuspected epidemiological events. Rev Inst Med Trop Sao Paulo, 61, e51, Merker, M., Kohl, T.A., Niemann, S., Supply, P. (2017) The Evolution of Strain Typing in the Mycobacterium tuberculosis Complex. Adv Exp Med Biol, 1019, 43-78). Другой подход заключается в использовании двух различных методов, основанных на ПЦР, одним из которых являлось бы сполиготипирование (Filliol, I., Ferdinand, S., Negroni, L., Sola, C, Rastogi, N. (2000) Molecular typing of Mycobacterium tuberculosis based on variable number of tandem DNA repeats used alone and in association with spoligotyping. J Clin Microbiol, 38,2520-2524). По своей разрешающей способности такой "тандем" приближается к традиционному фингерпринтингу по IS 6110. Ценность метода сполиготипирования заключается также в возможности одновременной детекции возбудителя, причем анализ может выполняться непосредственно из клинического образца без предварительного подращивания культуры, а также способность дискриминировать туберкулезные и бовисные штаммы, что представляет серьезные трудности при использовании общепринятых культуральных методов (Roring, S., Hughes, M.S., Skuce, R.A., Neill, S.D. (2000) Simultaneous detection and strain differentiation of Mycobacterium bovis directly from bovine tissue specimens by spoligotyping. Vet Microbiol, 74, 227-236).
Одним из недостатков предложенного в работе (Kamerbeek, J., Schouls, L., Kolk, A., Van Agterveld, M., Van Soolingen, D., Kuijper, S., Bunschoten, A., Molhuizen, H., Shaw, R., Goyal, M., Van Embden, J. (1997) Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology. J Clin Microbiol, 35, 907-914) варианта метода некоторыми исследователями указывается сравнительно большое время анализа - он занимает 1-2 рабочих дня, что на сегодняшнее время нельзя считать удовлетворительным.
Изобретение, описанное в патентной заявке RU 2237090 С1 «Способ генетического типирования штаммов микобактерий туберкулезного комплекса», связано с типированием по полиморфным локусам с применением мультиплексной системы праймеров. Способ позволяет устанавливать идентичность геномов микобактерий туберкулезного комплекса в эпидемиологических исследованиях и клинической медицине. Недостатком способа является сложность мультипраймерной схемы (пять пар праймеров, находящихся в общем реакционном объеме), возможные затруднения с интерпретацией результатов, а также недостаточная стандартизация по сравнению с другими методами типирования. Способ не использует биологический микрочип.
Изобретение RU 2689801 С1 «Способ детекции штаммов Mycobacterium bovis BCG в формате реального времени» описывает детекцию штаммов Mycobacterium bovis BCG путем лабораторного выявления геномной делеции RD1. Недостатком является невозможность типирования как Mycobacterium bovis, так и других представителей комплекса МБТ. Способ не использует биологический микрочип.
Изобретение CN 102094077 A "Method for real-time detection of beijing genotype mycobacterium tuberculosis" (КНР) относится к области медицинских тестов и обеспечивает набор для быстрого определения генотипа штамма Mycobacterium tuberculosis. Набор предназначен для детекции последовательности генетического элемента IS6110. Метод не позволяет проводить эпидемиологические исследования представителей микобактериального комплекса МБТ.
Применение метода "direct on-chip PCR" - непосредственная амплификация на биологическом микрочипе - позволяет решить проблему генотипирования параллельно с миниатюризацией процесса.
В настоящем изобретении описан метод сполиготипирования штаммов М tuberculosis и М. bovis (потенциально - и других представителей комплекса М. tuberculosis), использующий ПЦР на биологическом микрочипе с иммобилизованными праймерами, высокоспецифичньгми к DR-локусу (локус прямых повторов) микобактериального генома.
Раскрытие сущности изобретения
Изобретение, основанное на применение ПЦР на биологическом микрочипе (биочип), включает набор иммобилизованных в гелевых ячейках биочипа праймеров, специфичных к сорока трем спейсерным участкам DR-локуса, а также свободные праймеры, комплементарные прямому повтору: прямой DR-B1 и обратный флуоресцентно меченный DR-A1 (Фигура 1). Полный набор праймеров конструировали с учетом общепринятых требований (разброс расчетных температур плавления не более 2-3°С, отсутствие высокостабильных вторичных структур, высокая специфичность к выбранному региону). Кроме того, каждый иммобилизованный праймер проверяли на отсутствие дуплексов с праймерами DR-A1 и DR-B1 и анализировали на отсутствие сродства к другим локусам микобактериального и других геномов, последовательности которых доступны для BLAST-анализа. При расчете температур плавления дуплексов принимали во внимание влияние гелей биочипа на стабильность образующихся дуплексов. Поэтому реальная температура амплификации была понижена до 60°С.
В случае, когда длина спейсерного участка не позволяла выполнить последнее условие (обеспечение необходимой стабильности дуплекса), праймер конструировали таким образом, что его 5'-концевая область заходила в правую часть прямого повтора. Последовательности праймеров указаны в Перечне последовательностей. Иммобилизованные праймеры находятся в отдельных гелевых ячейках биочипа.
Структура самого биочипа определяется требованием миниатюризации, а именно уменьшением линейных размеров для удобства считывания получаемого ПЦР-профиля на портативном анализаторе биочипов. В результате биочип представляет собой упорядоченную матрицу 10x5 мм, элементами которой являются ячейки, содержащие праймеры, специфичные к сорока трем описанным в литературе и внесенным в базы данных прямым повторам. Сорок три олигонуклеотида расположены в порядке возрастания номера сверху вниз и слева направо, т.е. левый вертикальный столбец содержит первый десяток праймеров и т.д. (Фигура 2). Биочипы изготавливали по сополимеризационной технологии, когда праймер для иммобилизации вносится на стеклянную подложку биочипа вместе с раствором акриламида, из которого формируется ячейка.
Немаловажным этапом оптимизации метода явился выбор акриламидного геля, свойства которого позволяли проводить ПЦР с наибольшей эффективностью. Особые требования предъявляли к размеру пор в образующемся полимере, а также к его стабильности в процессе амплификации. Оптимизация условий используемой в методе ПЦР позволила получить интенсивные сигналы амплификации при высокой воспроизводимости результатов.
Процедура амплификации заканчивается инкубированием ПЦР-смеси 10 мин. при 72°С - для гибридизации полученных флуоресцентно меченных ПЦР-продуктов с экстендированными (достроенными) иммобилизованными праймерами (Фигура 1). Непосредственно после этого осуществляется отмывка биочипа для удаления неспецифичных сигналов. Это позволяет резко увеличить дискриминацию между сигналами ячеек с экстендированными (достроенными) праймерами и ячеек, содержащих праймеры, специфичные к спейсерам, отсутствующим в анализируемом образце. При этом сигналы последних сравнимы с сигналами пустых гелевых ячеек. Отношение положительных и отрицательных нормализованных сигналов превышает один порядок, что позволяет проводить визуальную интерпретацию результата (Фигура 2).
Детекция флуоресцентных сигналов ячеек осуществляется с использованием флуоресцентного анализатора биочипов. Анализ ПЦР-профиля проводят как визуально (Фигура 2), так и с помощью оригинального программного обеспечения "Analyze". В последнем случае появляется возможность количественной обработки интенсивностей флуоресцентных сигналов и нахождения значений нормализованных сигналов для автоматической программной интерпретации получаемых ПЦР-профилей. Результаты представляют в общепринятом для сполиготипирования виде - используя закрашенные области схемы для обозначения положительных сигналов (Таблица 1).
Заключительным этапом разработки метода явился сравнительный анализ результатов, полученных в ходе данной работы, и результатов стандартного сполиготипирования гибридизацией на мембранах. В Таблице 1 показаны профили, полученные стандартным и предлагаемым методами (обнаружено 100%-е совпадение профилей).
Краткое описание фигур и таблиц
Фигура 1. Схема сполиготипирования на биочипе (микрочипе, биологическом микрочипе). Прямые повторы идентичной последовательности, присутствующие в области прямых повторов (DR), амплифицируются с помощью праймеров DR-A1 и DR-B1. Праймер DR-A1 несет флуоресцентную метку и используется с 10-кратным молярным избытком по сравнению с праймеровм DR-B1; следовательно, ПЦР в растворе на биочипе дает преимущественно одноцепочечный флуоресцентно меченый продукт.Иммобилизованные праймеры, комплементарные 43 уникальным спейсерам, пронумерованы (для простоты показаны только 3 из них). В результате ПЦР образуются очень стабильные дуплексы между удлиненными иммобилизованными праймерами и флуоресцентно меченными одноцепочечными продуктами амплификации.
Фигура 2. Примеры профилей флуоресценции после проведения «ПЦР на чипе». Сполиготипы, полученные с использованием двух образцов ДНК, М. tuberculosis H37Rv (А) и М. bovis BCG М-34 (В). Раскладка биочипа (сетка) разделена на колонки по десять ячеек, номера колонок указаны вверху. Пятая колонка включает в себя последние три ячейки из 43.
Таблица 1. Результаты сполиготипирования, полученные традиционным (стандартным) методам гибридизации и методом ПЦР на биологическом микрочипе. Закрашенные серым цветом ячейки указывают на наличие соответствующих прямых повторов в ДНК исследуемого образца.
Осуществление изобретения
Изобретение нацелено на внутри- и межвидовое типирование бактериальных штаммов комплекса МБТ.
Ниже описаны основные этапы разработки способа, изготовления биологического микрочипа, синтеза олигонуклеотидных праймеров, а также осуществления способа.
Праймеры. Конструирование праймеров осуществляли с использованием программного обеспечения Oligo 5 (Molecular Biology Insights, Cascade, Colo.). Праймеры были синтезированы на синтезаторе ABI-394 DNA/RNA synthesizer (Applied Biosystems, Foster City, Calif.). Для иммобилизации праймеры модифицировали в автоматическом режиме присоединением 5'-Amino-Modifier С6 (Glen Research, Sterling, Va.). Праймеры были очищены методом ВЭЖХ на обращенно-фазовых колонках Cis Nucleosil (Sigma, St. Louis, Mo.). Праймер DR-A1 содержал флуоресцентную метку Texas Red® (Molecular Probes, Inc., Eugene, Oreg.), которая вводилась в соответствии с рекомендациями производителя.
DR-микрочип. Микрочипы изготавливались по методике сополимеризации, описанной ранее (Rubina, A.Y., Pan'kov, S.V., Dementieva, ЕЛ., Pen'kov, D.N., Butygin, A.V., Vasiliskov V.A., Chudinov, A.V., Mikheikin, A.L., Mikhailovich, V.M., Mirzabekov, A.D. (2004) Hydrogel drop microchips with immobilized DNA: properties and methods for large-scale production. Anal Biochem, 325, 92-106). Олигонуклеотиды вносились на стеклянную подложку вместе с остальными компонентами, формирующими ячейки. Сополимеризацию осуществляли методом фотополимеризации при λ = 260 нм в течение 30 мин. Диаметр полусферических ячеек равнялся 300 микрон при периоде 500 микрон. Размер активной области микрочипа составлял 4.8×2.3 мм.
ПЦР на DR-микрочипе. В 30 мкл реакционной смеси содержались: ПЦР-буфер (10 мМ KCl, 10 мМ Tris-HCl, рН 8.3), 3 мМ MgCl2, 200 мкМ каждого dNTP, 4 Ед фрагмента Стоффеля AmpliTaq ДНК-полимеразы (Perkin Elmer, USA). В ПЦР-смесь вносили праймеры: 2 пмоль прямого DR-B1 (SEQ ID NO: 1) и 20 пмоль обратного флуоресцентно меченного DR-A1 (SEQ ID NO: 2). Температурный режим реакции был, как описано ниже: 95°С - 3 мин., 40 циклов амплификации (30 с при 95°С, 40 с при 60°С и 40 с при 72°С), 72°С - 10 мин. ПЦР проводили в камере In situ Frame (Eppendorf Sci. Inc.) на амплификаторе TGradient (Whatman Biometra, Germany). После снятия реакционной камеры чип промывался трижды по 2 мин 0.1М раствором NaCl при 72°С, затем быстро ополаскивался бидистиллированной водой для удаления солевых разводов и высушивался в потоке воздуха.
Измерение флуоресцентного сигнала. Анализ ПЦР-профиля осуществляли на сухом микрочипе, используя портативный анализатор флуоресценции (чип-детектор). Изображение передавалось через ПЗС-камеру на компьютер для дальнейшей обработки. Измерение интенсивностей сигналов осуществляли программой "Analyze". Сохраняли как изображение ПЦР-профиля в формате pdf, так и файл с числовыми значениями сигналов.
Далее изобретение будет проиллюстрировано примерами, которые предназначены для обеспечения лучшего понимания сущности заявленного изобретения, но не должны рассматриваться как ограничивающие данное изобретение.
Пример 1. Специфичные к локусу прямых повторов (DR-локусу) олигонуклеотидные праймеры, используемые в способе.
Праймеры для проведения полимеразной цепной реакции на биологическом микрочипе синтезируют на автоматическом синтезаторе 394 DNA/RNA synthesizer (Applied Biosystems, США). Последовательности праймеров DR-A1 (SEQ ID NO: 1), DR-A1 (SEQ ID NO: 2), а также список иммобилизованных праймеров (SEQ ID NO: 3-45) представлен в Перечне последовательностей.
Пример 2. Проведение полимеразной цепной реакции для амплификации локуса прямых повторов (DR-локуса) с помощью полимеразной цепной реакции на биологическом микрочипе.
К 29 мкл реакционной смеси вносят 1 мкл образца геномной ДНК анализируемого бактериального штамма.
Мультиплексная ПЦР в иммобилизованной фазе.
Для проведения ПЦР используют Taq-полимеразу ("Thermo Scientific") в буфере, соответствующем набору для Taq-полимеразы.
1. К аликвоте смеси, содержащей реакционный буфер и природные dNTP (по 200 мкМ каждого) добавляют праймеры (10 мкМ обратного DR-A1 (SEQ ID NO: 1) и 1.0 мкМ прямого DR-B1 (SEQ ID NO: 2)).
2. Смесь помещают на биологический микрочип и герметизируют с помощью Frame-Seal 25 mkl ("Bio-Rad", США).
3. Проводят ПЦР на биологическом микрочипе с иммобилизованными 43 праймерами на ДНК-амплификаторе для in situ ПЦР TGradient Thermocycler ("Biometra", США) при следующих параметрах: 95°С в течение 3 мин (начальная денатурация); 36 циклов амплификации (20 с при 95°С, 30 с при 60°С и 40 с при 72°С) завершающая инкубация в течение 5 мин при 72°С.
4. Осуществляют промывку биологического микрочипа дистиллированной водой в течение 1 минуты при комнатной температуре, после чего биологический микрочип высушивают в струе воздуха.
5. Производят анализ полученного на биочипе ПЦР-профиля и интерпретацию результатов анализа с помощью "Чип-детектора".
Пример 3. Интерпретация результатов сполиготипирования (определения сполиготипа).
Считывание флуоресцентного сигнала с биочипа после удлинения иммобилизованных праймеров осуществляют с применением анализатора "Чип-детектор" (ИМБ, Россия). Интенсивность полученных сигналов определяют с помощью программного обеспечения "Analyse" ver. 1.0 (ИМБ, Россия) или "ImaGeWare" ver. 3.50 (ИМБ, Россия).
Интерпретацию наличия в образце того или иного набора спейсеров в локусе прямых повторов осуществляют визуально либо полуавтоматически с помощью программного обеспечения "Analyse" ver. 1.0 (ИМБ, Россия) или "ImaGeWare" ver. 3.50 (ИМБ, Россия).



--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="Способ сполиготипирования микобактерий туберкулезного
комплекса с использованием ДНК-амплификации в иммобилизованной фазе и
биологический микрочип для его осуществления.xml" softwareName="WIPO
Sequence" softwareVersion="2.2.0" productionDate="2023-03-19">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>28.02.2023</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт молекулярной биологии им. В.А.
Энгельгардта Российской академии наук (ИМБ РАН)</ApplicantName>
<ApplicantNameLatin>Engelhardt Institute of Molecular Biology,
Russian Academy of Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ сполиготипирования
микобактерий туберкулезного комплекса с использованием
ДНК-амплификации в иммобилизованной фазе и биологический микрочип для
его осуществления</InventionTitle>
<SequenceTotalQuantity>45</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q3">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>direct primer</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
прямой</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>DR-B1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccccgagaggggacggaaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q7">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>Texas Red</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q10">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>reverse fluorescently labeled
primer</INSDQualifier_value>
<NonEnglishQualifier_value>праймер обратный флуоресцентно
меченный</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>DR-A1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggggttttgggtctgacgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q14">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#1</INSDQualifier_value>
<NonEnglishQualifier_value>№1</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q16">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtcgccggttctggatcacgctcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q26">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#2</INSDQualifier_value>
<NonEnglishQualifier_value>№2</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q20">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcctcatgcttgggcgacagcttttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q25">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#3</INSDQualifier_value>
<NonEnglishQualifier_value>№3</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q24">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgccgtgcttccagtgatcgccttcta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q30">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#4</INSDQualifier_value>
<NonEnglishQualifier_value>№4</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q31">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcagtagcacgtcatacgccgaccaatcatc</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q35">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#5</INSDQualifier_value>
<NonEnglishQualifier_value>№5</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q36">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttttctgaccacttgtgcgggattagcggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q40">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#6</INSDQualifier_value>
<NonEnglishQualifier_value>№6</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q41">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccaatgcgtcgtcatttccggcttcaattt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q43">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q45">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#7</INSDQualifier_value>
<NonEnglishQualifier_value>#7</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q46">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgaggagagcgagtactcggggctgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q50">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q49">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#8</INSDQualifier_value>
<NonEnglishQualifier_value>№8</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q51">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaaccgcccccagcctcgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q53">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q55">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#9</INSDQualifier_value>
<NonEnglishQualifier_value>№9</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q56">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>NH2-group</NonEnglishQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcccatgtgctgacagcggattcgcat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q58">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q60">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q59">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#10</INSDQualifier_value>
<NonEnglishQualifier_value>№10</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q61">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgaaatacaggctccacgagaccaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q63">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q65">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q64">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#11</INSDQualifier_value>
<NonEnglishQualifier_value>№11</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q66">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagcaacggcggcggcaacctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q68">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q70">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q69">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#12</INSDQualifier_value>
<NonEnglishQualifier_value>№12</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q71">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcccggggagatgctgtccga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q73">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q75">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q74">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#13</INSDQualifier_value>
<NonEnglishQualifier_value>№13</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q76">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaccatcattgccattccctctccccac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q78">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q80">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q79">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#14</INSDQualifier_value>
<NonEnglishQualifier_value>№14</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q81">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgaaatacagccacgacacgaccaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q83">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q85">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q84">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#15</INSDQualifier_value>
<NonEnglishQualifier_value>№15</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q86">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggaaaccttgaataacgcgcagtgaatttcg</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q88">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q90">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q89">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#16</INSDQualifier_value>
<NonEnglishQualifier_value>№16</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q91">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgaacaggctccacgacacgaccaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q93">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q95">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q94">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#17</INSDQualifier_value>
<NonEnglishQualifier_value>№17</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q96">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggcccgcgcggatgactcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q98">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q100">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q99">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#18</INSDQualifier_value>
<NonEnglishQualifier_value>№18</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q101">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggcgagctgcagatggtccggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q103">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q105">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q104">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#19</INSDQualifier_value>
<NonEnglishQualifier_value>№19</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q106">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgctaactggcttggcgctgatcctggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q108">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q110">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q109">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#20</INSDQualifier_value>
<NonEnglishQualifier_value>№20</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q111">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccacatcgatttccttgacctcgccagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q113">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q115">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q114">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#21</INSDQualifier_value>
<NonEnglishQualifier_value>№21</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q116">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcatacaggctcccgacacgaccaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q118">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q120">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q119">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#22</INSDQualifier_value>
<NonEnglishQualifier_value>№22</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q121">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgtcaccgcagacggcacgattgag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q123">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q125">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q124">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#23</INSDQualifier_value>
<NonEnglishQualifier_value>№23</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q126">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcatcgctgatgcggtccagctcgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q128">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q130">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q129">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#24</INSDQualifier_value>
<NonEnglishQualifier_value>№24</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q131">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggaaatcaggctccacgcacgaccaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q133">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q135">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q134">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#25</INSDQualifier_value>
<NonEnglishQualifier_value>№25</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q136">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatcagcgaccaccgcaccctgtca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q138">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q140">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q139">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#26</INSDQualifier_value>
<NonEnglishQualifier_value>№26</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q141">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaaccttgaatacgcgcagtgaattcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q143">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q145">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q144">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#27</INSDQualifier_value>
<NonEnglishQualifier_value>№27</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q146">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggaaacttgaataagcgcagtgaatttcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q148">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q150">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q149">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#28</INSDQualifier_value>
<NonEnglishQualifier_value>№28</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q151">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgccccggcgtttagcgatcacaaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q153">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q155">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q154">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#29</INSDQualifier_value>
<NonEnglishQualifier_value>№29</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q156">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgaaatacaggctccacgacacgaccaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q158">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q160">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q159">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#30</INSDQualifier_value>
<NonEnglishQualifier_value>№30</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q161">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccccgcgcccttttccagcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q163">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q165">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q164">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#31</INSDQualifier_value>
<NonEnglishQualifier_value>№31</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q166">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggaccttgtaacgcgcagaatttcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q168">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q170">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q169">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#32</INSDQualifier_value>
<NonEnglishQualifier_value>№32</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q171">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatcactcccgaccaaataggtatcggcgtgttc</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q173">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q175">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q174">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#33</INSDQualifier_value>
<NonEnglishQualifier_value>№33</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q176">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacatgacgacggtgccgcacttgacgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q178">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q180">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q179">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#34</INSDQualifier_value>
<NonEnglishQualifier_value>№34</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q181">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtcacctcgcccacaccgtcgaagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q183">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q185">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q184">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#35</INSDQualifier_value>
<NonEnglishQualifier_value>№35</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q186">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagtccgtacgctcgaaacgcttccaacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q188">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q190">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q189">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#36</INSDQualifier_value>
<NonEnglishQualifier_value>№36</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q191">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgaaatccagcaccacatccgcagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q193">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q195">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q194">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#37</INSDQualifier_value>
<NonEnglishQualifier_value>№37</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q196">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgcgaactcgtccacagtccccct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q198">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q200">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q199">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#38</INSDQualifier_value>
<NonEnglishQualifier_value>№38</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q201">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtggatggcggatgcgttgtgcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q203">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q205">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q204">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#39</INSDQualifier_value>
<NonEnglishQualifier_value>№39</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q206">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacgatggccagtaaatcggcgtgggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q208">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q210">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q209">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#40</INSDQualifier_value>
<NonEnglishQualifier_value>№40</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q211">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccatctgtgcctcatacaggtccagtgccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q213">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q215">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q214">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#41</INSDQualifier_value>
<NonEnglishQualifier_value>№41</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q216">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgacggcaaggagctttccggcttctatcagg</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q218">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q220">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q219">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#42</INSDQualifier_value>
<NonEnglishQualifier_value>№42</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q221">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggacatggacgagcgcgactatcggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q223">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q225">
<INSDQualifier_name>product</INSDQualifier_name>
<INSDQualifier_value>immobilized primer</INSDQualifier_value>
<NonEnglishQualifier_value>иммобилизованный
праймер</NonEnglishQualifier_value>
</INSDQualifier>
<INSDQualifier id="q224">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>#43</INSDQualifier_value>
<NonEnglishQualifier_value>№43</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q226">
<INSDQualifier_name>function</INSDQualifier_name>
<INSDQualifier_value>NH2-group</INSDQualifier_value>
<NonEnglishQualifier_value>аминогруппа</NonEnglishQualifier_val
ue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggacgcagaatcgcaccgggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
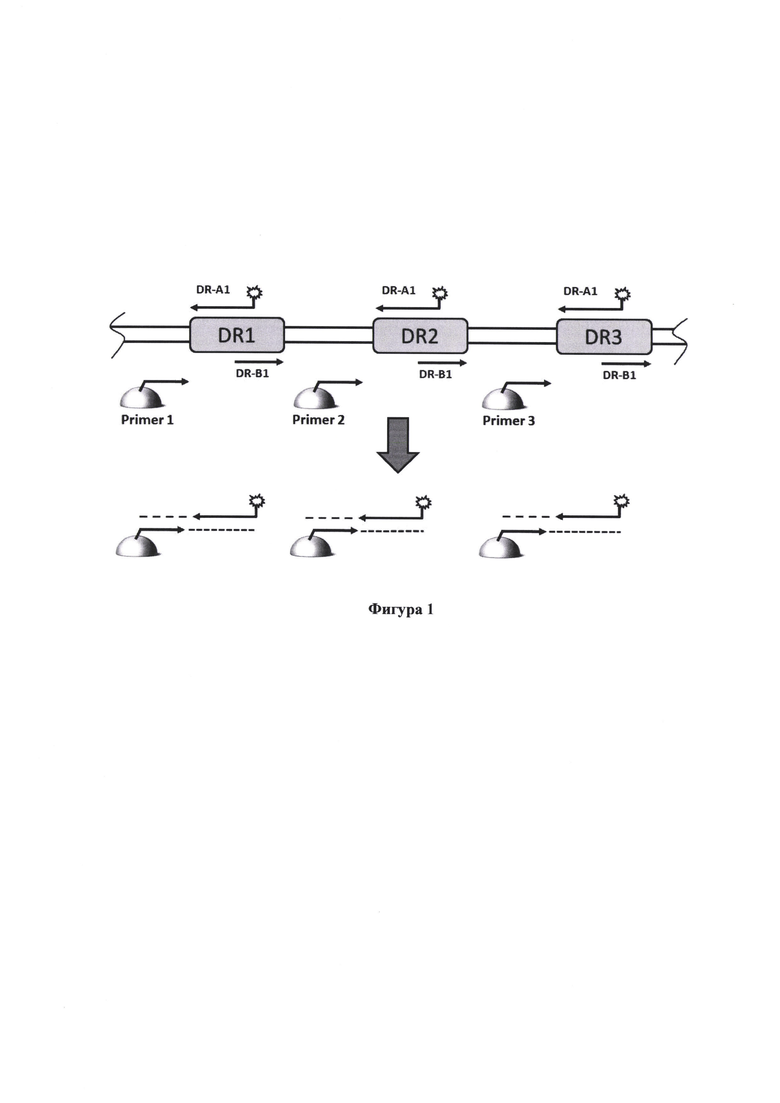

Изобретение относится к молекулярной биологии. Описан способ молекулярного генетического типирования Mycobacterium tuberculosis и Mycobacterium bovis, представляющий собой амплификацию локуса прямых повторов геномов Mycobacterium tuberculosis и Mycobacterium bovis с применением полимеразной цепной реакции на биологическом микрочипе с высокоспецифичными иммобилизованными праймерами, соответствующими сорока трем спейсерам DR-локуса. Также описан биологический микрочип, используемый в способе сполиготипирования методом полимеразной цепной реакции, с гидрогелевыми ячейками с иммобилизованными в них высокоспецифичными праймерами. Техническим результатом является реализация изобретением указанного назначения. 2 н.п. ф-лы, 2 ил., 1 табл., 3 пр.
1. Способ молекулярного генетического типирования Mycobacterium tuberculosis и Mycobacterium bovis, а именно сполиготипирование методом полимеразной цепной реакции на биологическом микрочипе, включающий:
а) амплификацию локуса прямых повторов геномов Mycobacterium tuberculosis и Mycobacterium bovis с применением полимеразной цепной реакции с помощью двух оригинальных олигонуклеотидных праймеров, находящихся в растворе над биологическим микрочипом, представленных последовательностями SEQ ID NO: 1, SEQ ID NO: 2, и сорока трех оригинальных олигонуклеотидных праймеров, иммобилизованных на биологическом микрочипе и представленных последовательностями SEQ ID NO: 3-45;
б) отмывку биологического микрочипа;
в) анализ картины сполиготипирования на биологическом микрочипе с помощью чип-детектора;
г) визуальную интерпретацию результатов анализа.
2. Биологический микрочип, используемый в способе сполиготипирования методом полимеразной цепной реакции на биологическом микрочипе, с гидрогелевыми ячейками с иммобилизованными в них высокоспецифичными праймерами, представленными последовательностями SEQ ID NO: 3-45.
| Bespyatykh JA, Zimenkov DV, Shitikov EA, Kulagina EV, Lapa SA, Gryadunov DA, Ilina EN, Govorun VM | |||
| Способ защиты переносных электрических установок от опасностей, связанных с заземлением одной из фаз | 1924 |
|
SU2014A1 |
| Spoligotyping of Mycobacterium tuberculosis complex isolates using hydrogel oligonucleotide microarrays | |||
| Прибор для получения стереоскопических впечатлений от двух изображений различного масштаба | 1917 |
|
SU26A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| СПОСОБ ВЫЯВЛЕНИЯ МИКОБАКТЕРИЙ ТУБЕРКУЛЕЗА ГЕНОТИПА BEIJING | 2008 |
|
RU2405836C2 |
| CN 108977503 A, 11.12.2018. | |||
Авторы
Даты
2023-11-21—Публикация
2023-04-10—Подача