Группа изобретений относится к области генной инженерии, а именно к числу средств - нуклеотидной последовательности, кодирующей фермент литиказу, и панели олигонуклеотидов для синтеза такой нуклеотидной последовательности, которые могут быть использованы для получения препарата фермента литиказы в виде рекомбинантного белка в клетках E.coli с высоким выходом фермента в растворимой форме.
Литиказа - фермент, относящийся к классу β-1,3-глюканаз (КФ 3.2.1.39) и катализирующий гидролиз глюканов, содержащих -1,3-связанные мономеры глюкозы. В-1,3-глюканазы вырабатываются бактериями и грибами как часть внеклеточного комплекса ферментов, которые, действуя синергетически, способны лизировать жизнеспособные дрожжевые клетки.
Клеточная стенка дрожжей представляет собой высокодинамичную структуру, которая отвечает за защиту клетки от быстрого изменения внешнего осмотического потенциала. Дрожжевая стенка состоит в основном из маннопротеина и β-1,3-глюканов с небольшим количеством ответвлений β-1,6-глюканов. Глюканы являются важными компонентами, ответственными за механическую прочность, форму и эластичность клеточной стенки. Благодаря этому клеточная стенка грибов и дрожжей обладает большой устойчивостью к большинству литических агентов (растворы детергентов, щелочей и хаотропных солей), что сильно затрудняет выделение ДНК из них. Выделение ДНК в достаточном количестве для своевременного выявления патогена методами амплификации нуклеиновых кислот и последующего назначения адекватной противогрибковой терапии является актуальной задачей.
Ряд различных микроорганизмов продуцирует внеклеточные ферменты, лизирующие дрожжи: Cellulosimicrobium cellulans (ранее Oerskovia xanthineolytica, также известная как Arthrobacter luteus), Cytophaga johnsonii., Rhizoctonia solani., Micromonospora chalcea, Lysobacter enzymogenes. Актиномицеты Cellulosimicrobium cellulans считаются основным источником дрожжевых литических ферментов, особенно эндо-β-1,3-глюканаз, протеаз и маннаназ. Примечательно, что несколько коммерчески доступных препаратов дрожжевых литических глюканаз, полученных из этого организма, а именно литиказа, зимолиаза и квантазим, широко используются для получения дрожжевых протопластов и выделения дрожжевой ДНК. Различные штаммы бактерий Cellulosimicrobium cellulans секретируют широкий спектр β-1,3-глюканаз с разными физико-химическими и гетерогенными свойствами. Следует отметить, что все выделенные формы β-1,3-глюканаз проявляли гидролитическую активность по отношению к β-глюканам (глюканазная активность), но лишь некоторые из них были способны индуцировать лизис жизнеспособных дрожжевых клеток (литическая активность). Всего выделяется 3 вида β-1,3-глюканаз из двух штаммов Cellulosimicrobium cellulans. Штамм АТСС 21606 Cellulosimicrobium cellulans продуцирует глюканазу массой 54,5 кДа, которая обладает высокой литической активностью в отношении клеточной стенки дрожжей. Штамм DSM 10297 Cellulosimicrobium cellulans продуцирует β-1,3-глюканазы BgI II (массой 40,8 кДа) и BgI IIa (массой 28,6 кДа), но только β-1,3-глюканаза BgI II обладает литической активностью. Это связано с тем, что у фермента BgI IIa отсутствует глюкан- или маннан-связывающий домен, в отличие от других β-1,3-глюканаз и протеаз, обладающих высокой литической активностью в отношении грибов и дрожжей. β-1,3-глюканазы BgI II и BgI IIa относятся к 16 семейству гликозидгидролаз, а β-1,3-глюканаза массой 54,5 кДа относится к 64 семейству гликозидгидролаз.
Из уровня техники известен способ получения β-1,3-глюканазы из Oerskovia xanthineolytica LL-G109. В 1990 году A.M. Ventom и J.A. Asenjo [Ventom А.М., Asenjo J.А. Purification of the major glucanase of Oerskovia xanthineolytica LL-G109 // Biotechnology techniques. - 1990. - T. 4. - C. 165-170., https://doi.org/10.1007/BF00222499] описали способ выделения литических эндоглюканаз, секретируемых Oerskovia xanthineolytica LL-G109 и оценили β-1,3-глюканазную активность полученных ферментов. Протокол очистки фермента был очень длительным и включал в себя 5 последовательных стадий: анионообменная хроматография (сорбент DEAE Sepharose), гидрофобная хроматография (сорбент Phenyl Sepharose), гель-фильтрация, гидрофобная хроматография (сорбент Phenyl Sepharose) и гель-фильтрация. Выход фермента составил 19% от исходного количества белка. Такая многостадийная очистка является дорогостоящей, трудоемкой и время затратной. Кроме того, выход фермента составил всего 1 мг ферментов с 1 литра культуральной среды.
Выделение фермента из организма-хозяина не является оптимальным подходом: каждый организм обладает индивидуальными особенностями и требует подбора специфических сред и условий культивирования, а выход целевого фермента как правило оказывается невысоким.
Из уровня техники также известен способ получения β-1,3-глюканазы из Oerskovia xanthineolytica в виде рекомбинантного фермента в клетках E.coli. В 1990 году S. Shen и соавторы выделили ген β-1,3-глюканазы из Oerskovia xanthineolytica и определили его нуклеотидную последовательность, а затем экспрессировали его в клетках Е. coli [Shen S. Н. et al. Primary sequence of the glucanase gene from Oerskovia xanthineolytica. Expression and purification of the enzyme from Escherichia coli // Journal of biological chemistry. - 1991. - T. 266. - №2. - С. 1058-1063., https://doi.org/10.1016/S0021-9258(17)35282-1]. Преимуществом такого способа получения фермента является использование хорошо изученных и простых систем экспрессии и методов клонирования. В частности, использовали экспрессионный вектор рОР95-15, экспрессию проводили в клетках DH5α. Белок был почищен за одну стадию методом ВЭЖХ на катионообменном сорбенте. Данные о выходе целевого белка представлены не были. В качестве недостатка такого подхода следует отметить использование хроматографии высокого давления (ВЭЖХ). Это довольно дорогой метод и оборудование для него имеется далеко не во всех лабораториях. Методы для очистки рекомбинантных белков, основанные на хроматографии низкого давления, используют чаще, эти методы легко масштабируются и доступны многим исследователям.
Из уровня техники также известен способ получения β-1,3-глюканазы в виде рекомбинантного фермента в клетках Е. coli [Salazar О. et al. Overproduction, purification, and characterization of β-1,3-glucanase type II in Escherichia coli // Protein expression and purification. - 2001. - T. 23. - №. 2. - C. 219-225., https://doi.org/10.1006/prep.2001.1497]. Был использован вектор pET20b (+), с помощью которого получали рекомбинантный белок, содержащий сигнальную последовательность pelB и последовательность из 6 гистидинов на С-конце, что позволяет использовать аффинную хроматографию для очистки. Экспрессию проводили в клетках BL21 (DE3). Несмотря на то, что рекомбинантный белок содержал полигистидиновый тэг, протокол очистки фермента состоял из 4 последовательных стадий: осаждение 80% сульфатом аммония, диализ, две стадии металл-аффинной хроматографии (сорбент Ni-NTA His⋅Bind). Выход фермента составил 0,8% от исходного количества белка. С 1 литра культуральной среды выход целевого фермента составил 70 мг. В результате авторами был получен активный фермент. Оценка глюканазной активности фермента проводилась по отношению к различным полисахаридным субстратам, литическая активность фермента по отношению к клеткам дрожжей или грибов не была продемонстрирована.
В связи с этим актуальной является задача в получении простых и эффективных решений, позволяющих получать экспрессионную конструкцию, кодирующую литиказу.
Технический результат достигается за счет использования уникальной нуклеотидной последовательности с оптимизированным кодонным составом, кодирующей литиказу, а также за счет синтеза панели олигонуклеотидов для сборки упомянутой нуклеотидной последовательности.
Заявляемый панель олигонуклеотидов разработана на основе анализа данных об аминокислотной последовательности фермента литиказы дикого типа: beta-1,3-glucanase (Cellulosimicrobium cellulans) GenBank: AAA25520.1, представленной в базе данных National Center for Biotechnology Information (NCBI) [https://www.ncbi.nlm.nih.gov/protein/AAA25520.1/]. Данная последовательность содержит как последовательность самого фермента литиказы (β-1,3-глюканазы) (37-548 а.о.), так и последовательность сигнального пептида (1-36 а.о.). Для получения рекомбинантного фермента в клетках E.coli последовательность сигнального пептида была удалена, а на С-конец аминокислотной последовательности добавлены шесть остатков гистидина для последующей очистки белка с помощью металл-афинной хроматографии. На основе разработанной аминокислотной последовательности рекомбинантного фермента SEQ ID NO: 2 получают нуклеотидную последовательность гена рекомбинантного фермента. Для этого на первом этапе проводят обратную трансляцию аминокислотной последовательности рекомбинантной литиказы SEQ ID NO: 2 с использованием соответствующего программного обеспечения, например, программы VectorNTI, а затем оптимизируют нуклеотидный состав гена с учетом частоты встречаемости кодонов для бактериальной системы экспрессии на основе клеток E.coli. На концах нуклеотидной последовательности Lyt-gene SEQ ID NO: 1 введены сайты рестрикции NcoI - на 5'-конце, и XhoI на 3'-конце, для последующего переклонирования в экспрессионный вектор pET20b (+).
Панель олигонуклеотидов для синтеза нуклеотидной последовательности SEQ ID NO: 1 in vitro разрабатывается с помощью программы [Owczarzy, R., et al. IDT SciTools: A Suite for Analysis and Design of Nucleic Acid Oligomers. / Owczarzy, R., et al. // Nucleic Acids Research. - 2008. - Vol. 36, no. Web Server, pp. W163-69, https://doi.org/10.1093/nar/gkn198.], реализованной в виде веб-сервиса (https://eu.idtdna.com/calc/analyzer). Последовательности синтезированных олигонуклеотидных праймеров приведены в таблице 1.

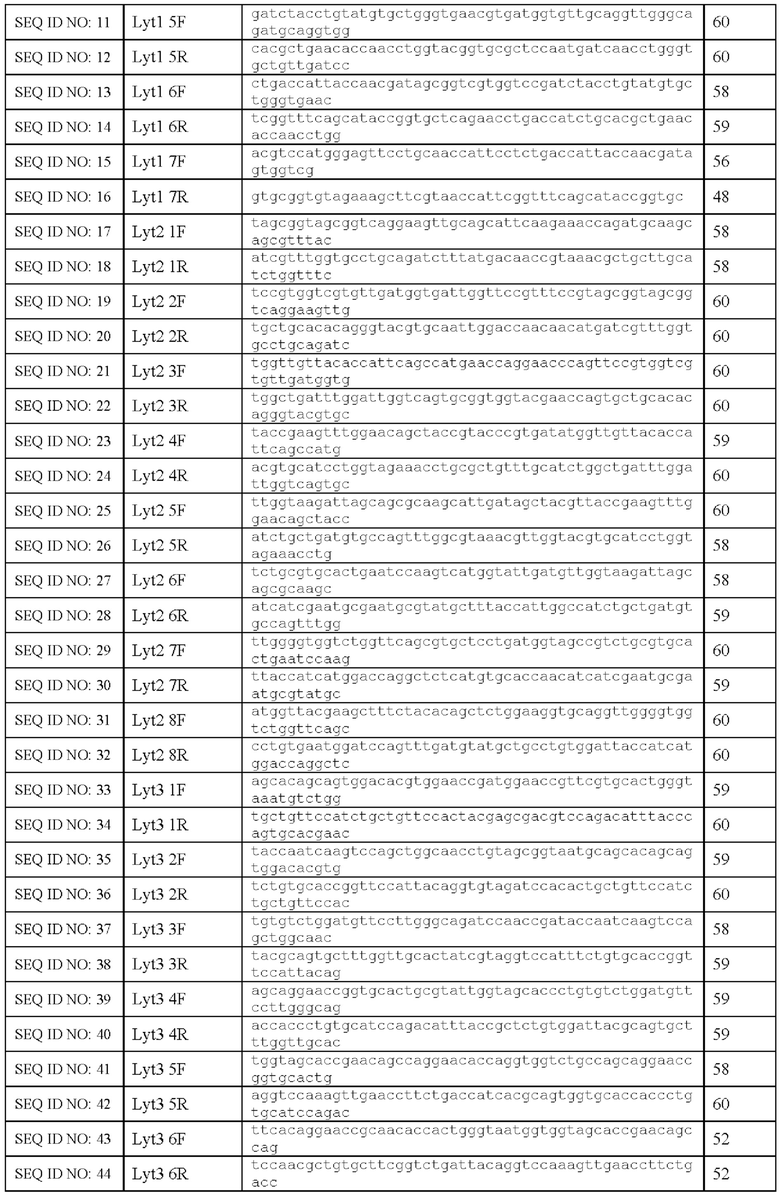

Нуклеотидную последовательность SEQ ID NO: 1 получают методом сборки из отдельных праймеров с помощью, так называемого «метода лесенки» [Xiong, A.S. А Simple, Rapid, High-Fidelity and Cost-Effective PCR-Based Two-Step DNA Synthesis Method for Long Gene Sequences./ Xiong, A.S. et al. // Nucleic Acids Research. - 2004. - Vol.32, no. 12, pp.e98-e98, https://doi.org/10.1093/nar/gnh094]. Синтез нуклеотидной последовательность SEQ ID NO: 1 проводят методом ПЦР, в два этапа, при этом на первом этапе проводят сборку трех фрагментов, где для сборки фрагмента 1 (Lyt1) используют праймеры SEQ ID NO: 3-16, для фрагмента 2 (Lyt2) - праймеры SEQ ID NO: 17-32, для фрагмента 3 (Lyt3) - праймеры SEQ ID NO: 33-46, а на втором этапе ПЦР полученные фрагменты сшивают при этом для сшивки фрагментов Lyt1 и Lyt2 используют праймеры SEQ ID NO: 3 (Lyt1 1F) и SEQ ID NO: 32 (Lyt2 8R) с получением фрагмента Lyt12, для сшивки фрагментов Lyt12 и Lyt3 используют праймеры SEQ ID NO: 3 (Lyt1 1F) и SEQ ID NO: 46 (Lyt3 7R) с получением фрагмента Lyt123.
Для получения нуклеотидной последовательности Lyt gene SEQ ID NO: 1 было необходимо введение стоп-кодона, для этого был проведен дизайн праймеров для мутагенеза (таблица 2).

Итоговая нуклеотидная последовательность: Lyt-gene SEQ ID NO: 1 дает возможность получения целевого фермента с полигистидиновым тэгом на С-конце SEQ ID NO: 2, что позволяет использовать высокоэффективный способ очистки белков методом аффинной хроматографии с использованием Ni-содержащих носителей, например, Ni-NTA агарозы.
Таким образом, при осуществлении заявляемого решения, в результате сборки синтетического гена из панели олигонуклеотидов SEQ ID NO: 3-46 получается уникальная нуклеотидная последовательность SEQ ID NO: 1, с кодонным составом, оптимизированным для экспрессии в клетках E.coli, кодирующая фермент литиказу (β-1,3-глюканазу). Заявляемое изобретение является результатом работы в рамках разработки ферментов, применяющихся в различных методах молекулярной диагностики, проделанной в ФБУН ЦНИИ Эпидемиологии Роспотребнадзора (Москва, Россия).
Реализация заявляемого изобретения поясняется следующими примерами:
Пример 1. Получение панели олигонуклеотидов и дизайн нуклеотидной последовательности литиказы из Cellulosimicrobium cellulans.
Панель олигонуклеотидов SEQ ID NO: 3-46 разработана на основе анализа данных об аминокислотной последовательности фермента литиказы дикого типа: beta-1,3-glucanase (Cellulosimicrobium cellulans) GenBank: AAA25520.1, представленной в базе данных National Center for Biotechnology Information (NCBI) [https://www.ncbi.nlm.nih.gov/protein/AAA25520.1/]. Данная последовательность содержит как последовательность самого фермента литиказы (β-1,3-глюканазы) (37-548 а.о.), так и последовательность сигнального пептида (1-36 а.о.). Для получения рекомбинантного фермента в клетках E.coli последовательность сигнального пептида была удалена, а на С-конец аминокислотной последовательности добавлены шесть остатков гистидина для последующей очистки белка с помощью металл-афинной хроматографии. На основе разработанной аминокислотной последовательности рекомбинантного фермента SEQ ID NO: 2 получили нуклеотидную последовательность гена рекомбинантного фермента. Для этого на первом этапе проводили обратную трансляцию аминокислотной последовательности рекомбинантной литиказы SEQ ID NO: 2 с использованием программы VectorNTI, а затем оптимизировали нуклеотидный состав гена с учетом частоты встречаемости кодонов для бактериальной системы экспрессии на основе клеток E.coli. На концах нуклеотидной последовательности Lyt-gene SEQ ID NO: 1 ввели сайты рестрикции NcoI - на 5'-конце, и XhoI - на 3'-конце, для последующего переклонирования в экспрессионный вектор pET20b (+).
Нуклеотидную последовательность SEQ ID NO: 1 получили методом сборки из отдельных праймеров с помощью, так называемого «метода лесенки» [Xiong, A.S. А Simple, Rapid, High-Fidelity and Cost-Effective PCR-Based Two-Step DNA Synthesis Method for Long Gene Sequences./ Xiong, A.S. et al. // Nucleic Acids Research. - 2004. - Vol. 32, no. 12, pp. e98-e98, https://doi.org/10.1093/nar/gnh094].
Панель олигонуклеотидов SEQ ID NO: 3-46 для синтеза нуклеотидной последовательности SEQ ID NO: 1 in vitro разрабатывали с помощью программы [Owczarzy, R., et al. IDT SciTools: A Suite for Analysis and Design of Nucleic Acid Oligomers. / Owczarzy, R., et al. // Nucleic Acids Research. - 2008. - Vol. 36, no. Web Server, pp. W163-69, https://doi.org/10.1093/nar/gkn198.], реализованной в виде веб-сервиса (https://eu.idtdna.com/calc/analyzer).
Для получения нуклеотидной последовательности Lyt gene SEQ ID NO: 1 было необходимо введение стоп-кодона, для этого был проведен дизайн праймеров для мутагенеза SEQ ID NO: 47-48.
Синтезированная нуклеотидная последовательность Lyt-gene SEQ ID NO: 1 дает возможность получения целевого фермента Lyt-CH с полигистидиновым тэгом на С-конце SEQ ID NO: 2.
Пример 2. Получение нуклеотидной последовательности литиказы из Cellulosimicrobium cellulans методом ПЦР и ее клонирование.
Нуклеотидная последовательность литиказы Lyt-gene SEQ ID NO: 1 была получена методом сборки из длинных перекрывающихся олигонуклеотидных праймеров SEQ ID NO: 3-46 (таблица 1). Синтез нуклеотидной последовательности SEQ ID NO: 1 проводили методом ПЦР с использованием высокоточной полимеразы Q5 High-Fidelity полимеразы фирмы New England Biolabs. На первом этапе проводили сборку трех фрагментов нуклеотидной последовательности, а затем фрагменты попарно сшивали между собой. Для сборки фрагмента 1 (Lyt1) использовали праймеры SEQ ID NO: 3-16, для фрагмента 2 (Lyt2) - праймеры SEQ ID NO: 17-32, для фрагмента 3 (Lyt3) - праймеры SEQ ID NO: 33-46.
Для сборки каждого фрагмента проводили ПЦР. Для фрагмента Lyt1 компоненты ПЦР смешивали следующим образом:
(a) 10,1 мкл реактива ddH2O (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия);
(b) 5 мкл реактива 5х Q5 Reaction Buffer (New England Biolabs, США);
(c) 5 мкл реактива 5х High GC Enhancer (New England Biolabs, США);
(d) 1 мкл 4.4 мМ dNTPs (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия);
(e) 1,4 мкл смеси праймеров, кроме концевых, каждый в концентрации 15 пмоль (на примере фргамента Lyt1 концевыми праймерами являются Lyt1 1F-NcoI и Lyt1 7R);
(f) 1 мкл концевого прямого праймера (Lyt1 1F-NcoI) в концентрации 30 пмоль;
(g) 1 мкл концевого обратного праймера (Lyt1 7R) в концентрации 30 пмоль;
(h) 0,5 мкл реактива Q5 High-Fidelity DNA Polymerase (New England Biolabs, США). Программа амплификации состояла из 24 циклов: денатурация при 98°С - 10 сек., отжиг - 15 сек. при 65°С, элонгация при 72°С - 30 сек, а также стадии предварительной денатурации при 98°С в течение 1 минуты и стадии финальной элонгации при 72°С в течение 2 минут.
Сборка других фрагментов Lyt2 и Lyt3 проводилась аналогичным образом. Полученные в результате проведения сборки ампликоны Lyt1, Lyt2 и Lyt3 очищали от побочных продуктов амплификации выделением из геля. На первом этапе проводили разделение фрагментов ДНК с помощью электрофореза в агарозном геле. Затем полоску геля, содержащую нужный продукт реакции вырезали. Выделение ДНК из геля проводили набором ZymocleanTM Gel DNA Recovery Kit фирмы «Zymo Research)) (США) согласно прилагаемой инструкции фирмы-производителя. Очищенный ампликон инкубировали 30 мин при 72°С в присутствии Taq-полимеразы и 0,44 мМ смеси дезоксинуклеотидов dNTPs для того, чтобы получить выступающие А-концы для последующего клонирования продукта в вектор pGEM-T (Promega). Целевой продукт лигировали в вектор pGEM-T (Promega). Наличие целевой последовательности и ее корректность подтверждали с помощью секвенирования методом Сэнгера.
По результатам секвенирования были отобраны клоны, не содержащие случайных мутаций, которые могут появляться за счет ошибок ДНК-полимеразы или неспецифического отжига праймеров.
Отобранные ампликоны сшивали попарно между собой с помощью ПЦР. Компоненты ПЦР смешиваются следующим образом (показано на примере сшивки фрагментов Lyt1 и Lyt2):
(a) 9,5 мкл реактива ddH2O (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия);
(b) 5 мкл реактива 5х Q5 Reaction Buffer (New England Biolabs, США);
(c) 5 мкл реактива 5х High GC Enhancer (New England Biolabs, США);
(d) 1 мкл 4.4 мМ dNTPs;
(e) 1 мкл очищенного ампликона одного сшиваемого фрагмента (Lyt1);
(f) 1 мкл очищенного ампликона второго сшиваемого фрагмента (Lyt2);
(g) 1 мкл концевого прямого праймера (Lyt1 1F-NcoI) в концентрации 30 пмоль;
(h) 1 мкл концевого обратного праймера (Lyt2 8R) в концентрации 30 пмоль;
(i) 0,5 мкл реактива Q5 High-Fidelity DNA Polymerase (New England Biolabs, США).
Программа амплификации состояла из 24 циклов: денатурация при 98°С - 10 сек., отжиг - 15 сек. при 57°С, элонгация при 72°С - 70 сек, а также стадии предварительной денатурации при 98°С в течение 1 минуты и стадии финальной элонгации при 72°С в течение 2 минут.
Для сшивки фрагментов Lyt1 и Lyt2 использовали праймеры Lyt1 1F (SEQ ID NO: 3) и Lyt2 8R (SEQ ID NO: 32) с получением фрагмента Lyt12. Для сшивки фрагментов Lyt12 и Lyt3 использовали праймеры Lyt1 1F (SEQ ID NO: 3) и Lyt3 7R (SEQ ID NO: 46) с получением фрагмента Lyt123.
Полученный в результате сшивки трех фрагментов ампликон очищали от побочных продуктов амплификации выделением из геля. На первом этапе проводили разделение фрагментов ДНК с помощью электрофореза в агарозном геле. Затем полоску геля, содержащую нужный продукт реакции, вырезали. Выделение ДНК из геля проводили набором ZymocleanTM Gel DNA Recovery Kit фирмы «Zymo Research)) (США) согласно прилагаемой инструкции фирмы-производителя. Очищенный ампликон инкубировали 30 мин при 72°С в присутствии Taq-полимеразы и 0,44 мМ смеси дезоксинуклеотидов dNTPs для того, чтобы получить выступающие А-концы для последующего клонирования продукта вектор pGEM-T (Promega). Целевой продукт лигировали в вектор pGEM-T (Promega). Наличие целевой последовательности и ее корректность подтверждали с помощью секвенирования методом Сэнгера.
Далее с использованием набора Q5 Site-Directed Mutagenesis Kit (New England Biolabs, США) на 3'-конец полученной нуклеотидной последовательность SEQ ID NO: 1 был введен стоп-кодон. Для этого использовали праймеры для мутагенеза SEQ ID NO 47-48, несущие соответствующую вставку. Реакцию мутагенеза проводили согласно инструкции производителя. Наличие целевой мутации подтверждали с помощью секвенирования методом Сэнгера.
Подготовленный описанным способом материал, содержащий уникальную нуклеотидную последовательность SEQ ID NO: 1, использовали для получения экспрессионной конструкции.
В результате получена плазмидная ДНК, содержащая последовательность гена, кодирующего литиказу из бактерий С.cellulans.
Пример 3. Получение экспрессионной конструкции p20Lyt_СН, кодирующей литиказу.
Для клонирования фрагмента, несущего целевой ген, проводили рестрикцию эндонуклеазами рестрикции NcoI и XhoI плазмиды pLyt_T, содержащей целевую последовательность гена, и вектора для экспрессии pET20b. Рестрикцию плазмидной ДНК проводили в течение 1-2 ч в соответствующем буфере при 37°С в суммарном объеме 20 мкл. Для гидролиза 1-2 мкл плазмидной ДНК (300 нг) брали 2-4 ед. эндонуклеазы рестрикции. Полноту протекания реакции контролировали, разделяя фрагменты в 1% агарозном геле. Затем проводили попарное лигирование целевой вставки и векторной ДНК и трансформацию плазмид в клетки E.coli XL1-blue. Единичные клоны, содержащие целевую вставку определяли методом ПЦР с универсальными праймерами T7-forward и Т7-reverse (Гентерра, Россия). Выделяли плазмидную ДНК и подтверждали корректность встраивания и наличие целевой последовательности с помощью секвенирования методом Сэнгера.
Таким образом, была получена экспрессионная конструкция, содержащая последовательность гена, кодирующего литиказу из бактерий С.Cellulans - p20Lyt_СН, содержащая нуклеотидную последовательность SEQ ID NO: 1.
Заявляемая группа изобретений позволяет получить нуклеотидную последовательность, кодирующую фермент литиказу, и панель олигонуклеотидов для синтеза такой нуклеотидной последовательности, которая может быть использована для получения экспрессионной конструкции, содержащей последовательность гена, кодирующего литиказу.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.2//EN" "ST26SequenceListing_V1_2.dtd">
<ST26SequenceListing dtdVersion="V1_2" fileName="Liticaza"
softwareName="WIPO Sequence" softwareVersion="1.0.0"
productionDate="2023-12-14">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>nn</ApplicationNumberText>
<FilingDate>2023-12-13</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>01</ApplicantFileReference>
<ApplicantName languageCode="ru">ФБУН ЦНИИ Эпидемиологии
Роспотребнадзора</ApplicantName>
<ApplicantNameLatin>FBUN CRIE</ApplicantNameLatin>
<InventionTitle languageCode="ru">Нуклеотидная последовательность,
кодирующая фермент литиказу, и панель олигонуклеотидов для получения
синтетической нуклеотидной последовательности гена
литиказы</InventionTitle>
<SequenceTotalQuantity>48</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>1632</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1632</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgaaatacctgctgccgaccgctgctgctggtctgctgctcctcgctg
cccagccggcgatggccatgggagttcctgcaaccattcctctgaccattaccaacgatagtggtcgtgg
tccgatctacctgtatgtgctgggtgaacgtgatggtgttgcaggttgggcagatgcaggtggaaccttc
catccttggcctggtggtgttggtcctgttcctgttccagctcctgatgcctctattgcaggtcctggtc
cgggtcagagcgttaccatccgtctgccgaaacttagtggtcgtgtttactacagctacggtcagaagat
gacatttcaaattgttctggatggacgtctggttcagccggcagttcagaacgatagcgatccgaaccgt
aacattctgttcaactggaccgaatacaccctgaacgatggtggtctgtggatcaacagcacccaggttg
atcattggagcgcaccgtaccaggttggtgttcagcgtgcagatggtcaggttctgagcaccggtatgct
gaaaccgaatggttacgaagctttctacacagctctggaaggtgcaggttggggtggtctggttcagcgt
gctcctgatggtagccgtctgcgtgcactgaatccaagtcatggtattgatgttggtaagattagcagcg
caagcattgatagctacgttaccgaagtttggaacagctaccgtacccgtgatatggttgttacaccatt
cagccatgaaccaggaacccagttccgtggtcgtgttgatggtgattggttccgtttccgtagcggtagc
ggtcaggaagttgcagcattcaagaaaccagatgcaagcagcgtttacggttgtcataaagatctgcagg
caccaaacgatcatgttgttggtccaattgcacgtaccctgtgtgcagcactggttcgtaccaccgcact
gaccaatccaaatcagccagatgcaaacagcgcaggtttctaccaggatgcacgtaccaacgtttacgcc
aaactggcacatcagcagatggccaatggtaaagcatacgcattcgcattcgatgatgttggtgcacatg
agagcctggtccatgatggtaatccacaggcagcatacatcaaactggatccattcacaggaaccgcaac
accactgggtaatggtggtagcaccgaacagccaggaacaccaggtggtctgccagcaggaaccggtgca
ctgcgtattggtagcaccctgtgtctggatgttccttgggcagatccaaccgataccaatcaagtccagc
tggcaacctgtagcggtaatgcagcacagcagtggacacgtggaaccgatggaaccgttcgtgcactggg
taaatgtctggacgtcgctcgtagtggaacagcagatggaacagcagtgtggatctacacctgtaatgga
accggtgcacagaaatggacctacgatagtgcaaccaaagcactgcgtaatccacagagcggtaaatgtc
tggatgcacagggtggtgcaccactgcgtgatggtcagaaggttcaactttggacctgtaatcagaccga
agcacagcgttggaccctgctcgagcaccaccaccaccaccac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1632</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SOURCE</INSDFeature_key>
<INSDFeature_location>1..1632</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>MOL_TYPE</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>ORGANISM</INSDQualifier_name>
<INSDQualifier_value>Cellulosimicrobium cellulans
</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>METLYSTYRLEULEUPROTHRALAALAALAGLYLEULEULEULEUALAA
LAGLNPROALAMETALAMETGLYVALPROALATHRILEPROLEUTHRILETHRASNASPSERGLYARGGL
YPROILETYRLEUTYRVALLEUGLYGLUARGASPGLYVALALAGLYTRPALAASPALAGLYGLYTHRPHE
HISPROTRPPROGLYGLYVALGLYPROVALPROVALPROALAPROASPALASERILEALAGLYPROGLYP
ROGLYGLNSERVALTHRILEARGLEUPROLYSLEUSERGLYARGVALTYRTYRSERTYRGLYGLNLYSME
TTHRPHEGLNILEVALLEUASPGLYARGLEUVALGLNPROALAVALGLNASNASPSERASPPROASNARG
ASNILELEUPHEASNTRPTHRGLUTYRTHRLEUASNASPGLYGLYLEUTRPILEASNSERTHRGLNVALA
SPHISTRPSERALAPROTYRGLNVALGLYVALGLNARGALAASPGLYGLNVALLEUSERTHRGLYMETLE
ULYSPROASNGLYTYRGLUALAPHETYRTHRALALEUGLUGLYALAGLYTRPGLYGLYLEUVALGLNARG
ALAPROASPGLYSERARGLEUARGALALEUASNPROSERHISGLYILEASPVALGLYLYSILESERSERA
LASERILEASPSERTYRVALTHRGLUVALTRPASNSERTYRARGTHRARGASPMETVALVALTHRPROPH
ESERHISGLUPROGLYTHRGLNPHEARGGLYARGVALASPGLYASPTRPPHEARGPHEARGSERGLYSER
GLYGLNGLUVALALAALAPHELYSLYSPROASPALASERSERVALTYRGLYCYSHISLYSASPLEUGLNA
LAPROASNASPHISVALVALGLYPROILEALAARGTHRLEUCYSALAALALEUVALARGTHRTHRALALE
UTHRASNPROASNGLNPROASPALAASNSERALAGLYPHETYRGLNASPALAARGTHRASNVALTYRALA
LYSLEUALAHISGLNGLNMETALAASNGLYLYSALATYRALAPHEALAPHEASPASPVALGLYALAHISG
LUSERLEUVALHISASPGLYASNPROGLNALAALATYRILELYSLEUASPPROPHETHRGLYTHRALATH
RPROLEUGLYASNGLYGLYSERTHRGLUGLNPROGLYTHRPROGLYGLYLEUPROALAGLYTHRGLYALA
LEUARGILEGLYSERTHRLEUCYSLEUASPVALPROTRPALAASPPROTHRASPTHRASNGLNVALGLNL
EUALATHRCYSSERGLYASNALAALAGLNGLNTRPTHRARGGLYTHRASPGLYTHRVALARGALALEUGL
YLYSCYSLEUASPVALALAARGSERGLYTHRALAASPGLYTHRALAVALTRPILETYRTHRCYSASNGLY
THRGLYALAGLNLYSTRPTHRTYRASPSERALATHRLYSALALEUARGASNPROGLNSERGLYLYSCYSL
EUASPALAGLNGLYGLYALAPROLEUARGASPGLYGLNLYSVALGLNLEUTRPTHRCYSASNGLNTHRGL
UALAGLNARGTRPTHRLEULEUGLUHISHISHISHISHISHIS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>56</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..56</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgtccatgggagttcctgcaaccattcctctgaccattaccaacgata
gtggtcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagacgtccatcaagaacaatttgaaatgtcatcttctgaccgtagctg
tagtaaacac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatgcctctattgcaggtcctggtccgggtcagagcgttaccatccgtc
tgccgaaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttcggatcgctatcgttctgaactgccggctgaaccagacgtccatcc
agaacgatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>57</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..57</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggtggtgttggtcctgttcctgttccagctcctgatgcctctattgc
aggtcctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagggtgtattcggtccagttgaacagaatgttacggttcggatcgcta
tcgttctgaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttgggcagatgcaggtggaaccttccatccttggcctggtggtgttgg
tcctgttcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caacctgggtgctgttgatccacagaccaccatcgttcagggtgtattc
ggtccagttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatctacctgtatgtgctgggtgaacgtgatggtgttgcaggttgggca
gatgcaggtgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacgctgaacaccaacctggtacggtgcgctccaatgatcaacctgggt
gctgttgatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgaccattaccaacgatagcggtcgtggtccgatctacctgtatgtgc
tgggtgaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcggtttcagcataccggtgctcagaacctgaccatctgcacgctgaac
accaacctgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>56</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..56</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgtccatgggagttcctgcaaccattcctctgaccattaccaacgata
gtggtcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgcggtgtagaaagcttcgtaaccattcggtttcagcataccggtgc<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tagcggtagcggtcaggaagttgcagcattcaagaaaccagatgcaagc
agcgtttac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcgtttggtgcctgcagatctttatgacaaccgtaaacgctgcttgca
tctggtttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccgtggtcgtgttgatggtgattggttccgtttccgtagcggtagcgg
tcaggaagttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgctgcacacagggtacgtgcaattggaccaacaacatgatcgtttggt
gcctgcagatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggttgttacaccattcagccatgaaccaggaacccagttccgtggtcg
tgttgatggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggctgatttggattggtcagtgcggtggtacgaaccagtgctgcacac
agggtacgtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taccgaagtttggaacagctaccgtacccgtgatatggttgttacacca
ttcagccatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgtgcatcctggtagaaacctgcgctgtttgcatctggctgatttgga
ttggtcagtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttggtaagattagcagcgcaagcattgatagctacgttaccgaagtttg
gaacagctacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atctgctgatgtgccagtttggcgtaaacgttggtacgtgcatcctggt
agaaacctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctgcgtgcactgaatccaagtcatggtattgatgttggtaagattagc
agcgcaagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcatcgaatgcgaatgcgtatgctttaccattggccatctgctgatgt
gccagtttgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttggggtggtctggttcagcgtgctcctgatggtagccgtctgcgtgca
ctgaatccaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttaccatcatggaccaggctctcatgtgcaccaacatcatcgaatgcga
atgcgtatgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggttacgaagctttctacacagctctggaaggtgcaggttggggtgg
tctggttcagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctgtgaatggatccagtttgatgtatgctgcctgtggattaccatcat
ggaccaggctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcacagcagtggacacgtggaaccgatggaaccgttcgtgcactgggt
aaatgtctgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgctgttccatctgctgttccactacgagcgacgtccagacatttaccc
agtgcacgaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taccaatcaagtccagctggcaacctgtagcggtaatgcagcacagcag
tggacacgtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctgtgcaccggttccattacaggtgtagatccacactgctgttccatc
tgctgttccac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtgtctggatgttccttgggcagatccaaccgataccaatcaagtcca
gctggcaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacgcagtgctttggttgcactatcgtaggtccatttctgtgcaccggt
tccattacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcaggaaccggtgcactgcgtattggtagcaccctgtgtctggatgtt
ccttgggcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accaccctgtgcatccagacatttaccgctctgtggattacgcagtgct
ttggttgcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggtagcaccgaacagccaggaacaccaggtggtctgccagcaggaacc
ggtgcactg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggtccaaagttgaaccttctgaccatcacgcagtggtgcaccaccctg
tgcatccagac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>52</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..52</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcacaggaaccgcaacaccactgggtaatggtggtagcaccgaacagc
cag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>52</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..52</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccaacgctgtgcttcggtctgattacaggtccaaagttgaaccttctg
acc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>38</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catcaaactggatccattcacaggaaccgcaacaccac</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>37</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgtctcgagcagggtccaacgctgtgcttcggtctg</INSDSeq_se
quence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgttggaccctgtaactcgagcaccacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtggtgctcgagttacagggtccaacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799413C1 |
| Способ определения генотипа ротавирусов первой и второй геногрупп и реассортантов между ними методом мультиплексной ПЦР | 2024 |
|
RU2833073C1 |
| НАБОР ПРАЙМЕРОВ ДЛЯ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799414C1 |
| СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2023 |
|
RU2816767C1 |
| НАБОР ОЛИГОНУКЛЕОТИДОВ И СПОСОБ ОБОГАЩЕНИЯ ГЕНОМНОЙ КДНК ВИРУСА КРЫМСКОЙ-КОНГО ГЕМОРРАГИЧЕСКОЙ ЛИХОРАДКИ ГЕНОТИПА ЕВРОПА-1 (V) МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2023 |
|
RU2839313C1 |
| Способ анализа крови ВИЧ-инфицированных пациентов | 2024 |
|
RU2830447C1 |
| Технология для детекции биозараженности нефтегазовых объектов | 2023 |
|
RU2812465C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА GBSSI У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2817377C1 |
| Нуклеотидная последовательность, кодирующая слитый белок, состоящий из функционального фрагмента человеческого IL-1RA и константной части тяжелой цепи человеческого IgG4 | 2023 |
|
RU2821896C1 |
| СПОСОБ ВЫЯВЛЕНИЯ СОБЫТИЙ РЕДАКТИРОВАНИЯ ГЕНА ISA1 У ЗЕРНОВЫХ КУЛЬТУР С ПОМОЩЬЮ НАБОРА ОЛИГОНУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ | 2023 |
|
RU2833963C1 |
Изобретение относится к области биотехнологии. Предложена панель олигонуклеотидов для получения гена литиказы с нуклеотидной последовательностью SEQ ID NO: 1, имеющая структуру олигонуклеотидных праймеров SEQ ID NO: 3-46, Ген литиказы, имеющий нуклеотидную последовательность, кодирующую фермент литиказу с SEQ ID NO: 1. Изобретение позволяет получать экспрессионную конструкцию, кодирующую литиказу, и получать литиказу в виде рекомбинантного белка в клетках Е.coli с высоким выходом фермента в растворимой форме. 2 н. и 3 з.п. ф-лы, 2 табл., 3 пр.
1. Панель олигонуклеотидов для получения гена литиказы с нуклеотидной последовательностью SEQ ID NO: 1, имеющая структуру олигонуклеотидных праймеров SEQ ID NO: 3-46.
2. Панель олигонуклеотидов по п. 1, где для создания олигонуклеотидных праймеров используют фрагмент гена литиказы дикого типа beta-1,3-glucanase Celluosimicrobium cellulans, при этом олигонуклеотидные праймеры обеспечивают синтез нуклеотидной последовательности гена литиказы.
3. Ген литиказы, имеющий нуклеотидную последовательность, кодирующую фермент литиказу с SEQ ID NO: 1.
4. Ген литиказы по п. 3, где нуклеотидную последовательность SEQ ID NO: 1 получают путем обратной трансляции аминокислотной последовательности рекомбинантной литиказы SEQ ID NO: 2, а на концах нуклеотидной последовательности SEQ ID NO: 1 вводят сайты рестрикции NcoI - на 5'-конце и XhoI на 3'-конце.
5. Ген литиказы по п. 3, синтез которого проводят с применением панели олигонуклеотидов SEQ ID NO: 3-46 по п. 1 методом ПЦР в два этапа, при этом на первом этапе проводят сборку трех фрагментов, где для сборки фрагмента 1 (Lyt1) используют праймеры SEQ ID NO: 3-16, для фрагмента 2 (Lyt2) - праймеры SEQ ID NO: 17-32, для фрагмента 3 (Lyt3) - праймеры SEQ ID NO: 33-46, а на втором этапе ПЦP полученные фрагменты сшивают, при этом для сшивки фрагментов Lyt1 и Lyt2 используют праймеры SEQ ID NO: 3 (Lyt1 1F) и SEQ ID NO: 32 (Lyt2 8R) с получением фрагмента Lyt12, для сшивки фрагментов Lyt12 и Lyt3 используют праймеры SEQ ID NO: 3 (Lyt1 1F) и SEQ ID NO: 46 (Lyt3 7R) с получением фрагмента Lyt123.
| Salazar О | |||
| et al | |||
| Overproduction, purification, and characterization of b-1,3-glucanase type II in Escherichia coli, Protein expression and purification, 2001, т | |||
| Прибор для равномерного смешения зерна и одновременного отбирания нескольких одинаковых по объему проб | 1921 |
|
SU23A1 |
| Прибор для записи звуковых волн | 1920 |
|
SU219A1 |
| Черкашина А.С | |||
| и др., Оптимизированная последовательность гена для получения рекомбинантной литиказы из бактерий | |||
Авторы
Даты
2024-09-04—Публикация
2023-12-18—Подача