Изобретение относится к области биотехнологии, молекулярно-генетической диагностики и может быть использовано для генотипирования человека по аллелям rs1052133-C и rs1052133-G гена OGG1, кодирующего фермент репарации окислительных повреждений ДНК 8-оксогуанин-ДНК-гликозилазу [Zhong Y, Zhang X, Feng R, Fan Y, Zhang Z, Zhang QW, Wan JB, Wang Y, Yu H, Li G. OGG1: An emerging multifunctional therapeutic target for the treatment of diseases caused by oxidative DNA damage. Med Res Rev. 2024 Aug 9. doi: 10.1002/med.22068]. Известно, что полиморфный вариант rs1052133 (C>G) гена OGG1 ассоциируется с высоким риском колоректального рака [Ghelmani Y, Asadian F, Antikchi MH, Dastgheib SA, Shaker SH, Jafari-Nedooshan J, Neamatzadeh H. Association Between the hOGG1 1245C>G (rs1052133) Polymorphism and Susceptibility to Colorectal Cancer: a Meta-analysis Based on 7010 Cases and 10,674 Controls. J Gastrointest Cancer. 2021 Jun;52(2):389-398. doi: 10.1007/s12029-020-00532-7], однако на сегодняшний день нет простого, быстрого и недорогого метода генотипирования человека по полиморфному локусу rs1052133 (C>G).
Методом, позволяющим осуществить определение нуклеотидной последовательности в локусе rs1052133 является секвенирование по Сэнгеру - метод, применяемый для детекции любых изменений нуклеотидной последовательности, разработанный Ф. Сэнгером в 1977 году и широко использующийся для изучения нуклеотидной последовательности ДНК человека [Sanger F. "Determination of nucleotide sequences in DNA". Biosci Rep. - 2004. - V. 24(4-5). - P.237-253]. Недостатками данного метода являются его трудоемкость, необходимость приобретения дорогостоящего оборудования и реактивов, значительно более долгое получение результатов и повышенные риски контаминации лаборатории продуктами полимеразно-цепной реакции (ПЦР) и реакций секвенирования.
Генотипирование rs1052133 гена OGG1 возможно методом секвенирования следующего поколения [Grada A, Weinbrecht K. Next-generation sequencing: methodology and application. J Invest Dermatol. 2013 Aug;133(8):e11. doi: 10.1038/jid.2013.248]. Недостатком метода является его высокая трудоемкость, длительность анализа и необходимость приобретения дорогостоящего секвенатора.
Генотипирование ДНК-полиморфизмов, включая rs1052133 гена OGG1, возможно осуществить методом матрично-активированной лазерной десорбционно-ионизационной масс-спектрометрии MALDI-TOF [Pusch W, Wurmbach JH, Thiele H, Kostrzewa M. MALDI-TOF mass spectrometry-based SNP genotyping. Pharmacogenomics. 2002 Jul;3(4):537-48. doi: 10.1517/14622416.3.4.537]. Недостатком метода является длительность анализа - не менее 8 часов, необходимость приобретения дорогостоящего геномного масс-спектрометра и набора чипов и реагентов.
Прототипом является метод анализа ДНК путем проведения полимеразно-цепной реакции в реальном времени в присутствии меченых флуорофорами, аллель-специфичных (TaqMan) зондов и праймеров по патенту на изобретение США №5538848 (Kenneth J. Livak, Susan J., A. Flood, Jeffrey Marmaro, Method for detecting nucleic acid amplification using self-quenching fluorescence probe, 23.07.1996). Недостатком прототипа является то, что для использования описанного в прототипе метода на практике, с целью исследования различных полиморфизмов, требуется разработка TaqMan зондов и праймеров для каждого отдельного исследуемого полиморфизма.
Технический результат заключается в разработке простого, быстрого и экономически выгодного способа генотипирования однонуклеотидного варианта rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени.
Технический результат достигается тем, что для генотипирования однонуклеотидного варианта rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени используются: прямой праймер rs1052133 5`-CTAGTCTCACCAGCCCTGAC-3` (SEQ ID NO 1), обратный праймер rs1052133 5`-GTGCCCCATCTAGCCTTCC-3` (SEQ ID NO 2), rs1052133-C-аллель-специфичный флуоресцентно-меченый зонд 5′-(FAM)CCGACCTGCGCCAATC(RTQ1)-3′ (SEQ ID NO 3), rs1052133-G-аллель-специфичный флуоресцентно-меченый зонд 5′-(ROX)CCGACCTGCGCCAATG(BHQ)-3′ (SEQ ID NO 4), при этом для гомозиготных по аллелю C образцов ДНК с генотипом rs1052133-C/C детектируется нарастание флуоресценции по каналу FAM, для гомозиготных по аллелю G образцов ДНК с генотипом rs1052133-G/G детектируется сигнал по каналу ROX, для гетерозиготных образцов с генотипом rs1052133-C/G наблюдается нарастание флуоресценции по обоим каналам детекции, а наличие двух красителей FAM и ROX позволяет определить присутствие каждого из исследуемых аллелей гена OGG1 в анализируемом образце ДНК и, соответственно, генотип человека.
Изобретение поясняется двумя фигурами. На фигуре 1 представлены нуклеотидные последовательности фрагмента гена OGG1 длиной 162 пары нуклеотидов, кодирующие аллели rs1052133-C и rs1052133-G гена OGG1. Жирным шрифтом и подчеркиванием отмечены последовательности праймеров, жирным шрифтом и курсивом - последовательности зондов.
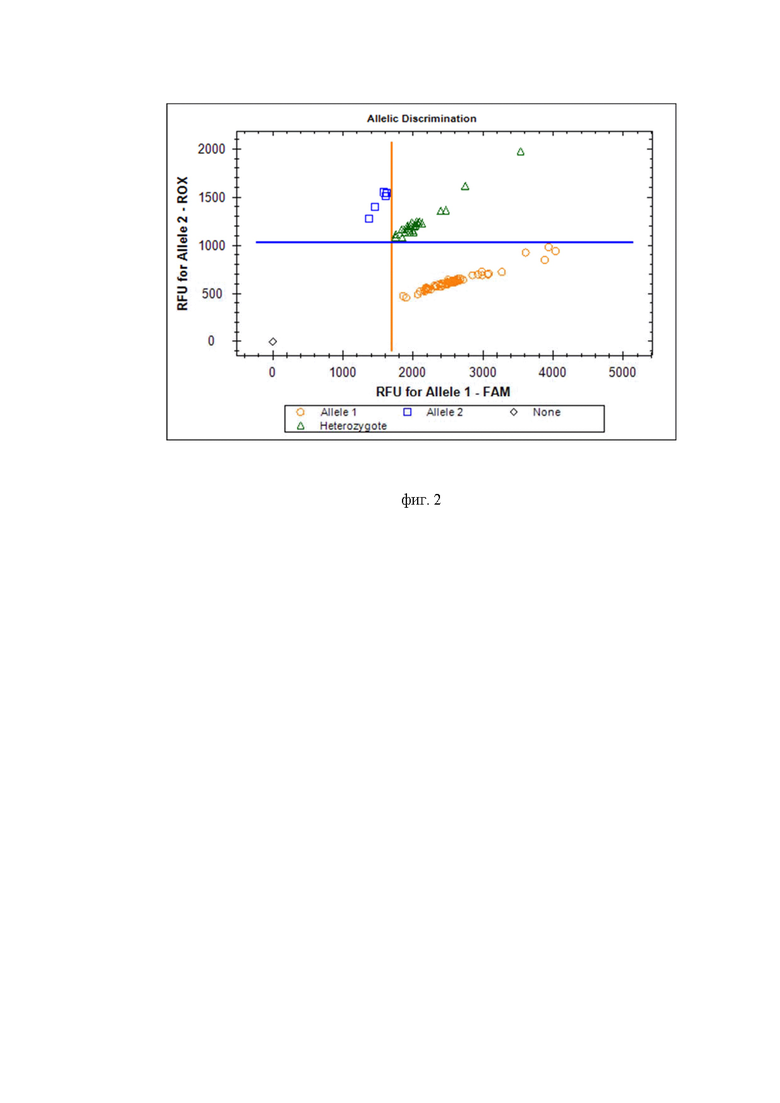
На фигуре 2 представлен пример детекции генотипов по локусу rs1052133 гена OGG1 при генотипировании методом полимеразно-цепной реакции в режиме реального времени: генотипы rs1052133-C/C показаны оранжевым цветом, генотипы rs1052133-C/G показаны зеленым цветом, генотипы rs1052133-G/G показаны синим цветом, черным ромбом показан отрицательный контроль.
Способ осуществляют следующим образом.
Для генотипирования однонуклеотидного варианта rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени использовали два общих для аллельных вариантов праймера, фланкирующих участок гена OGG1 длиной 162 пары нуклеотидов: прямой праймер rs1052133 5`-CTAGTCTCACCAGCCCTGAC-3` (SEQ ID NO 1), обратный праймер rs1052133 5`-GTGCCCCATCTAGCCTTCC-3` (SEQ ID NO 2), rs1052133-C-аллель-специфичный флуоресцентно-меченый зонд 5′-(FAM)CCGACCTGCGCCAATC(RTQ1)-3′ (SEQ ID NO 3), rs1052133-G-аллель-специфичный флуоресцентно-меченый зонд 5′-(ROX)CCGACCTGCGCCAATG(BHQ)-3′ (SEQ ID NO 4), подобранные на основе нуклеотидной последовательности гена OGG1 (https://www.ensembl.org/Homo_sapiens/Variation/Sequence?db=core;r=3:9756589-9757589;v=rs1052133;vdb=variation;vf=183299210), включающей в положении chr3:9757089 (GRCh38.p14) однонуклеотидный вариант rs1052133 (C>G). Для подбора праймеров использовали программу Primer3web version 4.1.0 (https://primer3.ut.ee/). Праймеры и зонды были синтезированы компанией «Синтол» (г. Москва). Прямой и обратный праймеры инициируют амплификацию участка гена OGG1, включающего локус rs1052133, длиной 162 пары нуклеотидов. Реакцию амплификации проводили в 13,25 мкл смеси ПЦР, содержащей 9,4 мкл ddH2O, 1,3 мкл раствора MgCl2 (массовая концентрация 2,5%), 1,3 мкл ПЦР-буфера, 0,2 мкл смеси дНТФ (концентрация 2 ммоль/л), 0,05 мкл раствора прямого и обратного праймера (концентрация 100 пкмоль/мкл), 0,025 мкл раствора каждого TaqMan-зонда (концентрация 100 пкмоль/мкл) и 0,11 мкл Taq ДНК-полимеразы (концентрация 5 Ед/мкл), 1 мкл образца ДНК (минимальная концентрация 10 нг/мкл). ПЦР проводили с помощью прибора CFX96 Real-Time System (Bio-Rad) при следующем режиме: 2 минуты при 50°С, 10 мин при 95°С, затем 38 циклов амплификации, включающей 15 секунд при 95°С и 30 секунд при t=56°С. Идентификация аллелей rs1052133-C и rs1052133-G гена OGG1 проводится на основании сравнения интенсивности флуоресценции красителей FAM и ROX, соответственно. Для определения генотипа человека используются конечные значения флуоресценции красителей FAM и ROX. Анализ результатов генотипирования проводили с помощью программного обеспечения для амплификатора CFX96 Real-Time System (Bio-Rad) версии Bio-Rad CFX Manager 2.1, которое представляет результаты генотипирования в виде распределения аллелей (фиг. 2). Для гомозиготных по аллелю C образцов ДНК с генотипом rs1052133-C/C детектируется нарастание флуоресценции по каналу FAM, для гомозиготных по аллелю G образцов ДНК с генотипом rs1052133-G/G детектируется сигнал по каналу ROX, для гетерозиготных образцов с генотипом rs1052133-C/G наблюдается нарастание флуоресценции по обоим каналам детекции, а наличие двух красителей FAM и ROX позволяет определить присутствие каждого из исследуемых аллелей гена OGG1 в анализируемом образце ДНК и, соответственно, генотип человека.
Разработанный способ был апробирован на 95 образцах ДНК человека из коллекции биобанка НИИ генетической и молекулярной эпидемиологии ФГБОУ ВО КГМУ Минздрава России. По результатам генотипирования 67,4% людей являлись гомозиготами по аллелю C (генотип rs1052133-C/C), 5,2% людей - гомозиготами по аллелю G (rs1052133-G/G), 27,4% - гетерозиготами (генотип rs1052133-C/G). Валидацию способа проводили с помощью матрично-активированной лазерной десорбционно-ионизационной масс-спектрометрии MALDI-TOF [Pusch W, Wurmbach JH, Thiele H, Kostrzewa M. MALDI-TOF mass spectrometry-based SNP genotyping. Pharmacogenomics. 2002 Jul;3(4):537-48. doi: 10.1517/14622416.3.4.537] на геномном масс-спектрометре MassArray Analyzer 4 (Agena Bioscience). Результаты обоих способов генотипирования полностью совпали, однако патентуемый способ генотипирования однонуклеотидного варианта rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени позволяет значительно (до 1 часа) сократить время проведения анализа по сравнению с методом генотипирования на основе MALDI-TOF (8 часов).
Примеры конкретного выполнения способа.
Пример 1. Образец ДНК DM523 из биобанка НИИ генетической и молекулярной эпидемиологии КГМУ, был подвергнут генотипированию по однонуклеотидному варианту rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени. К 1 мкл раствора ДНК с концентрацией 10 нг/мкл добавляли 13,25 мкл смеси ПЦР, содержащей 9,4 мкл ddH2O, 1,3 мкл раствора MgCl2 (массовая концентрация 2,5%), 1,3 мкл ПЦР-буфера, 0,2 мкл смеси дНТФ (концентрация 2 ммоль/л), 0,05 мкл раствора прямого и обратного праймера (концентрация 100 пкмоль/мкл), 0,025 мкл раствора каждого TaqMan-зонда (концентрация 100 пкмоль/мкл) и 0,11 мкл Taq ДНК-полимеразы (концентрация 5 Ед/мкл), 1 мкл образца ДНК (минимальная концентрация 10 нг/мкл). ПЦР проводили с помощью прибора CFX96 Real-Time System (Bio-Rad) при следующем режиме: 2 минуты при 50°С, 10 мин при 95°С, затем 38 циклов амплификации, включающей 15 секунд при 95°С и 30 секунд при t=56°С. При регистрации сигнала флуоресценции наблюдали экспоненциальный рост флуоресценции по ROX между 18 и 38 циклами, что свидетельствует о наличии в образце только аллелей rs1052133-G, что соответствует генотипу rs1052133-G/G OGG1.
Пример 2. Образец ДНК DM483 из биобанка НИИ генетической и молекулярной эпидемиологии КГМУ, был подвергнут генотипированию по однонуклеотидному варианту rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени. К 1 мкл раствора ДНК с концентрацией 10 нг/мкл добавляли 13,25 мкл смеси ПЦР, содержащей 9,4 мкл ddH2O, 1,3 мкл раствора MgCl2 (массовая концентрация 2,5%), 1,3 мкл ПЦР-буфера, 0,2 мкл смеси дНТФ (концентрация 2 ммоль/л), 0,05 мкл раствора прямого и обратного праймера (концентрация 100 пкмоль/мкл), 0,025 мкл раствора каждого TaqMan-зонда (концентрация 100 пкмоль/мкл) и 0,11 мкл Taq ДНК-полимеразы (концентрация 5 Ед/мкл), 1 мкл образца ДНК (минимальная концентрация 10 нг/мкл). ПЦР проводили с помощью прибора CFX96 Real-Time System (Bio-Rad) при следующем режиме: 2 минуты при 50°С, 10 мин при 95°С, затем 38 циклов амплификации, включающей 15 секунд при 95°С и 30 секунд при t=56°С. При регистрации сигнала флуоресценции наблюдали экспоненциальный рост флуоресценции по FAM и ROX между 18 и 38 циклами, что свидетельствует о наличии в образце и аллеля rs1052133-C, и аллеля rs1052133-G, что соответствует гетерозиготному генотипу rs1052133-C/G OGG1.
Пример 3. Образец ДНК DM482 из биобанка НИИ генетической и молекулярной эпидемиологии КГМУ, был подвергнут генотипированию по однонуклеотидному варианту rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени. К 1 мкл раствора ДНК с концентрацией 10 нг/мкл добавляли 13,25 мкл смеси ПЦР, содержащей 9,4 мкл ddH2O, 1,3 мкл раствора MgCl2 (массовая концентрация 2,5%), 1,3 мкл ПЦР-буфера, 0,2 мкл смеси дНТФ (концентрация 2 ммоль/л), 0,05 мкл раствора прямого и обратного праймера (концентрация 100 пкмоль/мкл), 0,025 мкл раствора каждого TaqMan-зонда (концентрация 100 пкмоль/мкл) и 0,11 мкл Taq ДНК-полимеразы (концентрация 5 Ед/мкл), 1 мкл образца ДНК (минимальная концентрация 10 нг/мкл). ПЦР проводили с помощью прибора CFX96 Real-Time System (Bio-Rad) при следующем режиме: 2 минуты при 50°С, 10 мин при 95°С, затем 38 циклов амплификации, включающей 15 секунд при 95°С и 30 секунд при t=56°С. При регистрации сигнала флуоресценции наблюдали экспоненциальный рост флуоресценции по FAM между 18 и 38 циклами, что свидетельствует о наличии в образце только аллелей rs1052133-C, что соответствует генотипу rs1052133-C/C OGG1.
Результаты генотипирования трех образцов ДНК DM482, DM483, DM523 по однонуклеотидному варианту rs1052133 (C>G) гена OGG1 с помощью матрично-активированной лазерной десорбционно-ионизационной масс-спектрометрии MALDI-TOF на геномном масс-спектрометре MassArray Analyzer 4 (Agena Bioscience) полностью совпали с результатами генотипирования методом полимеразно-цепной реакции в режиме реального времени.
Таким образом, разработан простой, быстрый и экономически выгодный способ генотипирования однонуклеотидного варианта rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени, который позволяет до 1 часа сократить время проведения анализа и дает возможность провести генотипирование по указанному полиморфному варианту в лаборатории, укомплектованной стандартным оборудованием - амплификатором для проведения ПЦР.
--->
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd"
PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN">
<ST26SequenceListing productionDate="2024-09-03"
softwareVersion="2.3.0" softwareName="WIPO Sequence" fileName="Способ
генотипирования однонуклеотидного варианта rs1052133 гена OGG1
человека методом полимеразно-цепной реакции в режиме реального
времени.xml" dtdVersion="V1_3">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText/>
<FilingDate>2024-09-03</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1934</ApplicantFileReference>
<ApplicantName languageCode="ru">федеральное государственное
бюджетное образовательное учреждение высшего образования «Курский
государственный медицинский университет» Министерства здравоохранения
Российской Федерации</ApplicantName>
<ApplicantNameLatin>Kursk State Medical
University</ApplicantNameLatin>
<InventorName languageCode="ru">Азарова Юлия
Эдуардовна</InventorName>
<InventorNameLatin>Azarova Yulia Eduardovna</InventorNameLatin>
<InventionTitle languageCode="ru">Способ генотипирования
однонуклеотидного варианта rs1052133 (C>G) гена OGG1 человека методом
полимеразно-цепной реакции в режиме реального
времени</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctagtctcaccagccctgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgccccatctagccttcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccgacctgcgccaatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccgacctgcgccaatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---

Изобретение относится к области биотехнологии. Описан способ генотипирования однонуклеотидного варианта rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени, в котором согласно изобретению применяются разработанные олигонуклеотидные праймеры. Технический результат заключается в разработке простого и быстрого способа генотипирования однонуклеотидного варианта rs1052133 (C>G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени. 2 ил., 3 пр.
Способ генотипирования однонуклеотидного варианта rs1052133 (C˃G) гена OGG1 человека методом полимеразно-цепной реакции в режиме реального времени, отличающийся тем, что используются: прямой праймер rs1052133 5′-CTAGTCTCACCAGCCCTGAC-3′ (SEQ ID NO 1), обратный праймер rs1052133 5′-GTGCCCCATCTAGCCTTCC-3′ (SEQ ID NO 2), rs1052133-C-аллель-специфичный флуоресцентно-меченый зонд 5′-(FAM)CCGACCTGCGCCAATC(RTQ1)-3′ (SEQ ID NO 3), rs1052133-G-аллель-специфичный флуоресцентно-меченый зонд 5′-(ROX)CCGACCTGCGCCAATG(BHQ)-3′ (SEQ ID NO 4), при этом для гомозиготных по аллелю C образцов ДНК с генотипом rs1052133-C/C детектируется нарастание флуоресценции по каналу FAM, для гомозиготных по аллелю G образцов ДНК с генотипом rs1052133-G/G детектируется сигнал по каналу ROX, для гетерозиготных образцов с генотипом rs1052133-C/G наблюдается нарастание флуоресценции по обоим каналам детекции, а наличие двух красителей FAM и ROX позволяет определить присутствие каждого из исследуемых аллелей гена OGG1 в анализируемом образце ДНК и, соответственно, генотип человека.
| СПОСОБ ЛЕЧЕНИЯ ПАРЕЗА ЖЕЛУДОЧНО-КИШЕЧНОГО ТРАКТА | 2005 |
|
RU2308994C2 |
| Способ генотипирования гена TLR6 по полиморфизму rs5743810 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805863C1 |
| Mao G | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Nucleic acids research, 2007, V.35 (8), P | |||
| ТЕЛЕФОННОЕ УСТРОЙСТВО | 1924 |
|
SU2759A1 |
Авторы
Даты
2025-02-25—Публикация
2024-09-12—Подача