Область техники
[0001]
Настоящее изобретение относится к мутантному белку F RSV и к его применению.
Предшествующий уровень техники
[0002]
Инфекция, вызываемая респираторно-синцитиальным вирусом (RSV), представляет собой респираторную инфекцию, распространенную во всем мире. Дети в возрасте до 5 лет подвержены высокому риску обострения заболевания. Инфекция RSV приводит к воспалению нижних дыхательных путей более чем у 30 миллионов человек ежегодно, и более 3 миллионов человек ежегодно нуждаются в госпитализации. Поэтому, в настоящее время было предпринято много усилий по разработке вакцины для профилактики инфекции RSV. В прошлом, в 1965-66 гг., клинические испытания (испытуемыми были младенцы) проводились с использованием вируса, инактивированного формалином, в качестве вакцинного антигена. Однако, вместо снижения заболеваемости инфекционными болезнями, это привело к 16-кратному увеличению числа госпитализаций по поводу RSV-инфекции. Одной из причин является то, что во время инактивирующей обработки, F-антиген, который является одним из поверхностных антигенов вируса, денатурируется, и индуцируются только антитела с низкой активностью нейтрализации RSV, и, таким образом, иммунный ответ подавляется. Хотя с тех пор было предпринято множество попыток разработать вакцину, однако, эффективная вакцина все же не была получена.
[0003]
В соответствии с этим было проведено исследование с использованием сывороток здоровых взрослых людей, и было отмечено, что у здоровых взрослых редко наблюдалось тяжелое заражение RSV. В результате исследования было обнаружено, что сыворотки здоровых взрослых обладают высокой способностью к нейтрализации вирусной инфекции, и что эффект нейтрализации инфекции обусловлен антителом против F-белка RSV (Непатентный документ 1). Также были проведены исследования F-белка RSV, и было обнаружено, что зрелый F-белок имеет метастабильную конформацию перед слиянием (которая может называться в настоящей заявке «пре-F» или «пре-белком типа F») и наиболее стабильную конформацию после слияния (которая может называться здесь «пост-F» или «пост-белком типа F»), и, из-за своей нестабильности, конформация до слияния имеет тенденцию к раннему рефолдингу в конформацию после слияния в общем растворе вакцинной композиции.
Кроме того, поскольку структура пре-F, стабильное получение которой было трудно достижимо, стала более ясной (Непатентный документ 2), то было обнаружено, что пре-белок типа F образует тример, и что некоторые из эпитопов (эпитопы Φ, I, II, III, IV, V и т.п.), присутствующие в пре-белке типа F, теряются из-за структурного изменения в пост-F.
[0004]
В исследовании, в котором более подробно была изучена сыворотка взрослого человека, стало ясно, что среди антител против белка F, содержащихся в сыворотке, антитела с высокой способностью нейтрализовать вирусную инфекцию в основном представляют собой антитела, которые специфически связываются с эпитопом Φ и с эпитопом V, присутствующими в пре-белке типа F, и эти антитела специфически распознают пре-белок типа F, но не связываются с пост-F, и что указанная выше сыворотка также содержит антитело, которое также связывается с пост-F, но обладает низкой способностью нейтрализовать вирусную инфекцию (Непатентный документ 3). Кроме того, младенец с недостаточным «созреванием» иммунной системы подвержен высокому риску обострения симптомов во время инфекции. Однако, это вызвано тем, что у младенца, который был инфицирован в первый раз или имеет низкую частоту инфицирования, наблюдается небольшая способность индуцировать иммунитет, нацеленный на эпитоп Φ или эпитоп V. Эффективное индуцирование антител, обладающих высокой способностью нейтрализовать вирусную инфекцию в антигенной вакцине, имеет важное значение, и считается, что идеальным вакцинным антигеном является антиген, который может предпочтительно индуцировать антитела против эпитопа Φ или эпитопа V, обладающие нейтрализующей способностью.
[0005]
Были предприняты попытки стабилизировать пре-белок типа F, имеющий эпитоп Φ или эпитоп V, путем мутации или т.п., которые вводят искусственную дисульфидную связь в белок F RSV. Так, например, имеются сообщения о мутанте DS-Cav1 (Патентный документ 1, Непатентный документ 5, Непатентный документ 6) или о других мутантах (Патентный документ 2). Однако, стабилизация пре-белка типа F была недостаточной, и исследования в этой области все еще продолжаются.
Родственные документы
[Патентный документ]
[0006]
[Патентный документ 1] Международная публикация № 2017/172890.
[Патентный документ 2] Международная публикация № 2017/109629.
[Непатентные документы]
[0007]
[Непатентный документ 1] Ngwuta JO et al., Science Translational Medicine 2015; 309: 309ra162.
[Непатентный документ 2] Jason S. McLellan et al., Science 2013; 340: 1113.
[Непатентный документ 3] Morgan SA Gilman et al., 2016, Science Immunology, 1 (6), pii: eaaj1879.
[Непатентный документ 4] Eileen Goodwin et al., Immunity 2018; 48 (2): 339.
[Непатентный документ 5] Jason S. McLellan et al., Science 2013; 342:592.
[Непатентный документ 6] M. Gordon Joyce et al., Nature structural and molecular biology 2016; 23 (9): 811.
[Описание сущности изобретения]
[Проблемы, которые могут быть решены с помощью настоящего изобретения]
[0008]
Настоящее изобретение относится к разработке способа эффективного индуцирования антитела, обладающего высокой способностью нейтрализовать инфекцию RSV.
Средства решения этих проблем
[0009]
Авторами настоящего изобретения было обнаружено, что мутантный белок F RSV, включающий специфическую аминокислотную мутацию, эффективно индуцирует антитело, обладающее высокой способностью нейтрализовать инфекцию RSV, и на этой основе было разработано настоящее изобретение.
Настоящее изобретение включает:
[0010]
[1] Мутантный белок F RSV, имеющий мутацию, где мутация представляет собой замену лейцина, соответствующего лейцину в положении 141 аминокислотной последовательности SEQ ID NO: 1, или лейцина, соответствующего лейцину в положении 142, на цистеин, и замену лейцина, соответствующего лейцину в положении 373, на цистеин, с последующим образованием дисульфидной связи между цистеинами.
[2] Мутантный белок F RSV согласно [1], где мутантный белок F RSV происходит от RSV подтипа A или RSV подтипа B.
[3] Мутантный белок F RSV согласно [2], где RSV подтипа A представляет собой штамм RSV A2 или штамм RSV с длинной цепью.
[4] Мутантный белок F RSV согласно [2], где RSV подтипа B представляет собой штамм RSV 18537.
[5] Мутантный белок F RSV согласно любому из [1]-[4], обладающий способностью индуцировать антитело против пре-белка типа F RSV, и содержащий аминокислотную последовательность, где в аминокислотной последовательности, которая на 85% или более идентична SEQ ID NO: 2, лейцин, соответствующий лейцину в положении 141, или лейцин, соответствующий лейцину в положении 142, и лейцин, соответствующий лейцину в положении 373 аминокислотной последовательности SEQ ID NO: 1, заменены цистеинами с образованием дисульфидной связи между цистеинами.
[6] Мутантный белок F RSV согласно любому из [1]-[4], где, кроме того, аминокислота, образующая сайт распознавания фурином, который присутствует на С-концевой стороне области pep27, заменена таким образом, что сайт распознавания фурином не распознается фурином.
[7] Мутантный белок F RSV согласно [6], где аминокислота, образующая сайт распознавания фурином, представляет собой аминокислоту, выбранную из группы, состоящей из аргинина, соответствующего аргинину в положении 133, аргинина, соответствующего аргинину в положении 135, и аргинина, соответствующего аргинину в положении 136 аминокислотной последовательности SEQ ID NO: 1, и такая аминокислота заменена неосновной аминокислотой.
[8] Мутантный белок F RSV согласно [6] или [7], обладающий способностью индуцировать антитело против пре-белка типа F RSV, и содержащий аминокислотную последовательность, где в аминокислотной последовательности, которая на 85% или более идентична SEQ ID NO: 3, лейцин, соответствующий лейцину в положении 141, или лейцин, соответствующий лейцину в положении 142, и лейцин, соответствующий лейцину в положении 373 аминокислотной последовательности SEQ ID NO: 1, заменены цистеинами и, кроме того, аминокислота, выбранная из группы, состоящей из аргинина, соответствующего аргинину в положении 133, аргинина, соответствующего аргинину в положении 135, и аргинина, соответствующего аргинину в положении 136 аминокислотной последовательности SEQ ID NO: 1, заменена неосновной аминокислотой, где между цистеинами образуется дисульфидная связь.
[9] Мутантный белок F RSV согласно [7] или [8], где неосновная аминокислота представляет собой аспарагин.
[10] Мутантный белок F RSV согласно любому из [1]-[9], где, кроме того, треонин, соответствующий треонину в положении 189 аминокислотной последовательности SEQ ID NO: 1, и/или серин, соответствующий серину в положении 190, заменены гидрофобными аминокислотами.
[11] Мутантный белок F RSV согласно [10], где каждая гидрофобная аминокислота независимо выбрана из группы, состоящей из валина, изолейцина и лейцина.
[12] Мутантный белок F RSV согласно любому из [1]-[11], где, кроме того, лизин, соответствующий лизину в положении 42 аминокислотной последовательности SEQ ID NO: 1, заменен аргинином, и/или валин, соответствующий валину в положении 384, заменен треонином.
[13] Мутантный белок F RSV, имеющий мутацию, где мутация представляет собой замену глутаминовой кислоты, соответствующей глутаминовой кислоте в положении 60 аминокислотной последовательности SEQ ID NO: 1, некислотной аминокислотой.
[14] Мутантный белок F RSV согласно [13], где некислотная аминокислота выбрана из группы, состоящей из метионина, фенилаланина, лейцина, треонина и серина.
[15] Гибридный белок, содержащий: мутантный белок F RSV согласно любому из [1]-[14]; и домен мультимеризации, слитый с С-концом мутантного белка F RSV.
[16] Гибридный белок согласно [15], где домен мультимеризации представляет собой домен фолдона.
[17] Гибридный белок согласно [16], содержащий аминокислотную последовательность любой из SEQ ID NO: 10-13.
[18] Мультимер мутантного белка F RSV согласно любому из [1]-[14], где слитый белок согласно любому из [15]-[17] связан посредством домена мультимеризации.
[19] Мультимер согласно [18], являющийся тримером.
[20] Продукт в виде частиц мутантного белка F RSV согласно любому из [1] - [14] или мультимера согласно любому из [15] - [17], где мутантный белок F RSV или слитый белок содержит домен, образующий частицы, два или более мутантных белка или мультимера F RSV агрегируются посредством домена, образующего частицу, с образованием частицы, и этот образующий частицы домен расположен в положении, более близком к эпитопу I, чем к эпитопу Φ, на стерической структуре мутантного белка или мультимера F RSV.
[21] Продукт в виде частиц согласно [20], где два или более мутантных белков F RSV или слитых белков для получения продукта в виде частиц, в которых домен, образующий частицы, связан с С-концом мутантного белка F RSV или слитого белка, агрегируются посредством домена, образующего частицы.
[22] Продукт в виде частиц согласно [20] или [21], где мультимер представляет собой тример, состоящий из слитого белка согласно [16].
[23] Продукт в виде частиц согласно [22], где образующий частицы домен представляет собой домен Fc, состоящий из аминокислотной последовательности SEQ ID NO: 30, и домен, образующий частицы, агрегируют путем связывания с доменом Z белка А, иммобилизованного на VLP, полученной из модифицированного антигена HBs.
[24] Продукт в виде частиц согласно [22], где образующий частицы домен представляет собой пептид, состоящий из аминокислотной последовательности SEQ ID NO: 42.
[25] Гибридный белок для получения продукта в виде частиц, содержащий: мутантный белок F RSV согласно любому из [1]-[14] или слитый белок согласно любому из [15]-[17], и образующий частицы домен, слитый с его С-концом.
[26] Слитый белок для получения продукта в виде частиц согласно [25], где домен, образующий частицы, представляет собой домен Fc, состоящий из аминокислотной последовательности SEQ ID NO: 30.
[27] Слитый белок для получения продукта в виде частиц согласно [25], где образующий частицы домен представляет собой пептид, состоящий из аминокислотной последовательности SEQ ID NO: 42.
[28] Полинуклеотид, кодирующий мутантный белок F RSV согласно любому из [1]-[14], слитый белок согласно любому из [15]-[17] или слитый белок для получения продукта в виде частиц согласно любому из [25]-[27].
[29] Экспрессионный кластер, содержащий полинуклеотид согласно [28].
[30] Клетка-хозяин, содержащая экспрессионный кластер согласно [29].
[31] Иммуноген, содержащий: мутантный белок F RSV согласно любому из [1]-[14], слитый белок согласно любому из [15]-[17], мультимер согласно любому из [18]-[19] или продукт в виде частиц согласно любому из [20]-[24].
[32] Фармацевтическая композиция, содержащая экспрессионный кластер согласно [29] или иммуноген согласно [31].
[33] Вакцина RSV, содержащая экспрессионный кластер согласно [29] или иммуноген согласно [31].
[34] Фармацевтическая композиция согласно [32] или вакцина RSV согласно [33], предназначенные для введения человеку.
Эффект изобретения
[0011]
В соответствии с настоящим изобретением может быть разработан способ эффективного индуцирования антитела, обладающего высокой способностью нейтрализовать инфекцию RSV. В качестве пре-белка типа F может служить мутантный белок F RSV, обладающий значительно более высокой стабильностью по сравнению с обычным белком. Мутантный белок F RSV согласно изобретению обладает эффектом индуцирования группы антител, которые, как считается, обладают высокой способностью нейтрализовать вирус, по сравнению с группой антител, которые, как считается, в меньшей степени способствуют нейтрализации вируса, особенно у человека (здесь и далее, этот эффект упоминается как эффект преимущественного индуцирования группы антител, которые, как считается, обладают высокой способностью нейтрализовать вирус).
Краткое описание чертежей
[0012]
[Фиг. 1] График, иллюстрирующий стабильность при хранении эпитопа Φ в Примере 1 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 2] График, иллюстрирующий стабильность при хранении эпитопа Φ в Примере 2 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 3] График, иллюстрирующий тримеризацию в Примере 2 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 4] График, иллюстрирующий стабильность при хранении эпитопа Φ в Примере 3 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 5] График, иллюстрирующий тримеризацию в Примере 3 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 6] График, иллюстрирующий уровень экспрессии в Примере 3 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 7] График, иллюстрирующий стабильность при хранении эпитопа Φ в Примере 4 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 8] График, иллюстрирующий тримеризацию в Примере 4 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 9] График, иллюстрирующий стабильность при хранении эпитопа Φ в Примере 5 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 10] График, иллюстрирующий тримеризацию в Примере 5 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 11] График, иллюстрирующий свойство распознавания эпитопа Φ в Примере 5 в соответствии с одним вариантом осуществления настоящего изобретения.
[Фиг. 12] График, иллюстрирующий результаты тестов на адсорбцию антител, нейтрализующих вирус, в сыворотках человека в Примере 5 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 13] График, иллюстрирующий результаты тестов на адсорбцию антител, нейтрализующих вирус, в сыворотках человека в Примере 5 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 14] График, иллюстрирующий результаты тестов на антитела, индуцированные мышиным иммунитетом в Примере 5, согласно одному варианту осуществления настоящего изобретения.
[Фиг. 15] График, иллюстрирующий стабильность при хранении эпитопа Φ в Примере 6 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 16] График, иллюстрирующий тримеризацию в Примере 6 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 17] График, иллюстрирующий свойство распознавания эпитопа I в Примере 6 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 18] График, иллюстрирующий результаты тестов на антитела, индуцированные мышиным иммунитетом в Примере 6, согласно одному варианту осуществления настоящего изобретения.
[Фиг. 19] График, иллюстрирующий стабильность при хранении эпитопа Φ в Примере 7 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 20] График, иллюстрирующий тримеризацию в Примере 7 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 21] График, иллюстрирующий свойство распознавания эпитопа I в Примере 7 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 22] График, иллюстрирующий стабильность при хранении эпитопа Φ в Примере 7 согласно одному варианту осуществления настоящего изобретения (анализ на встраивание линкера).
[Фиг. 23] График, иллюстрирующий тримеризацию в Примере 7 согласно одному варианту осуществления настоящего изобретения (анализ на встраивание линкера).
[Фиг. 24] График, иллюстрирующий свойство распознавания эпитопа I в Примере 7 согласно одному варианту осуществления настоящего изобретения (анализ на встраивание линкера).
[Фиг. 25] График, показывающий диаметры частиц в Примере 7 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 26] График, иллюстрирующий результаты электрофореза в полиакриламидном геле в Примере 8 согласно одному варианту осуществления настоящего изобретения (часть 1).
[Фиг. 27] График, иллюстрирующий результаты электрофореза в полиакриламидном геле в Примере 8 согласно одному варианту осуществления настоящего изобретения (часть 2).
[Фиг. 28] График, иллюстрирующий стабильность при хранении эпитопа Φ в Примере 8 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 29] График, иллюстрирующий тримеризацию в Примере 8 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 30] График, иллюстрирующий свойство распознавания эпитопа I в Примере 8 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 31] График, показывающий диаметры частиц в Примере 8 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 32] График, иллюстрирующий результаты тестов на антитела, индуцированные мышиным иммунитетом в Примере 8, согласно одному варианту осуществления настоящего изобретения.
[Фиг. 33] График, иллюстрирующий результаты электрофореза в полиакриламидном геле в Примере 9 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 34] График, иллюстрирующий стабильность при хранении эпитопа Φ в Примере 9 согласно одному варианту осуществления настоящего изобретения.
[Фиг. 35] График, иллюстрирующий свойство распознавания эпитопа I в Примере 9 согласно одному варианту осуществления настоящего изобретения.
Способ осуществления изобретения
[0013]
Один вариант осуществления настоящего изобретения представляет собой мутантный белок F RSV, имеющий мутацию, где мутацией является замена лейцина, соответствующего лейцину в положении 141 аминокислотной последовательности SEQ ID NO: 1, или лейцина, соответствующего лейцину в положении 142, на цистеин, и замена лейцина, соответствующего лейцину в положении 373, на цистеин, и где между цистеинами образуется дисульфидная связь.
[0014]
Природный белок F RSV сначала экспрессируется в виде полипептида F0 (предшественника). Полипептид F0 подвергается процессингу после расщепления сигнала миграции эндоплазматического ретикулума внутриклеточной фуриноподобной протеазой в двух сайтах с образованием полипептида F1, полипептида F2 и полипептида Pep27. В данном случае, полипептид Pep27 вырезается и, таким образом, не образует часть зрелого белка F. Полипептид F2 происходит от N-концевой боковой части полипептида F0 и связан с полипептидом F1 двумя дисульфидными связями. Полипептид F1 происходит от С-концевой боковой части полипептида F0 и иммобилизует зрелый белок F на мембране посредством трансмембранного домена. Три протомера гетеродимера F2-F1 образуют зрелый белок F в виде тримера, который представляет собой пре-белок типа F, выполняющий функцию слияния мембраны вируса и мембраны клетки-мишени.
[0015]
Мутантный белок F RSV согласно изобретению включает полипептид F2 и полипептид F1 с N-концевой стороны. В качестве полипептида F1 предпочтительно используют полипептид F1, который не включает трансмембранную область и внутриклеточную область (далее он может называться полипептидом F1 (внеклеточной областью)). Кроме того, мутантный белок F RSV согласно изобретению может включать полипептид Pep27 на N-концевой стороне полипептида F1 в результате удаления одного из сайтов распознавания фуриноподобной протеазой посредством мутации, которая будет описана ниже. Кроме того, могут быть также включены сигнальная последовательность на N-концевой стороне полипептида F2, линкер между полипептидом F1 и полипептидом F2 и метка, используемая для очистки, или т.п. на С-концевой стороне. Кроме того, мутантный белок F RSV согласно изобретению предпочтительно образует тример.
[0016]
Мутантный белок F RSV согласно изобретению предпочтительно получают из RSV подтипа A или RSV подтипа B.
Конкретные примеры RSV подтипа A включают штамм RSV A2, штамм RSV с длинной цепью, штамм RSV S2 и штамм линии 19 RSV, и среди них предпочтительным является штамм RSV A2 или штамм RSV с длинной цепью.
Конкретные примеры RSV подтипа B включают штамм RSV 18537, штамм RSV B1 и штамм RSV 9320, и среди них предпочтительным является штамм RSV 18537.
[0017]
Аминокислотная последовательность SEQ ID NO: 1 представляет собой аминокислотную последовательность полипептида F0 белка F штамма RSV A2. В связи с этим описаны структурные элементы полипептида F0.
В SEQ ID NO: 1, аминокислотная последовательность сигнальной последовательности представляет собой аминокислотную последовательность в положениях 1-25.
В SEQ ID NO: 1, аминокислотная последовательность полипептида F2 представляет собой аминокислотную последовательность в положениях 26-109.
В SEQ ID NO: 1, аминокислотная последовательность области pep27 представляет собой аминокислотную последовательность в положениях 110-136.
В SEQ ID NO: 1, аминокислотная последовательность полипептида F1 (внеклеточная область) представляет собой аминокислотную последовательность в положениях 137-513.
В SEQ ID NO: 1, аминокислотная последовательность полипептида F1 (трансмембранная область и внутриклеточная область) представляет собой аминокислотную последовательность в положениях 514-574. Аминокислотная последовательность полипептида F1 (трансмембранная область и внутриклеточная область) может представлять собой последовательность, которая на 85% или более, на 90% или на 95% или более идентична аминокислотной последовательностью в положениях 514-574 SEQ ID NO: 1.
[0018]
Белок F RSV может включать полипептид F2 и полипептид F1 (внеклеточную область). Примером белка F RSV, который включает полипептид F2 и полипептид F1 (внеклеточную область), является аминокислотная последовательность SEQ ID NO: 2, которая включает, в указанном порядке, аминокислотную последовательность в положениях 26-109 SEQ ID NO: 1 и аминокислотную последовательность в положениях 137-513.
Белок F RSV может включать полипептид F2 и полипептид F1 (внеклеточную область), с которым связана область pep27. Примером белка F RSV, который включает, в указанном порядке, полипептид F2 и полипептид F1 (внеклеточную область), с которым связана область pep27, является аминокислотная последовательность SEQ ID NO: 3, которая включает аминокислотную последовательность в положениях 26-513 SEQ ID NO: 1.
Эти полипептиды могут представлять собой отдельные полипептидные цепи, и предпочтительно они связаны дисульфидными связями. То есть, белок F RSV может представлять собой комплекс таких полипептидов.
[0019]
Мутация, при которой лейцин заменен цистеином
Мутантный белок F RSV согласно изобретению включает мутацию, а именно, замену лейцина, соответствующего лейцину в положении 141 аминокислотной последовательности SEQ ID NO: 1, или лейцина, соответствующего лейцину в положении 142, на цистеин, и замену лейцина, соответствующего лейцину в положении 373, на цистеин. В мутантном белке F RSV, имеющем вышеуказанную аминокислотную мутацию, в дополнение к встречающейся в природе дисульфидной связи между введенными цистеинами образуется новая дисульфидная связь, и, таким образом, достигается эффект повышения стабильности при хранении (сохранении пре-типа) эпитопа Ф. Для образования тримера, в частности, лейцин, соответствующий лейцину в положении 141 SEQ ID NO: 1, заменяют цистеином, и лейцин, соответствующий лейцину в положении 373, заменяют цистеином.
[0020]
В настоящем описании, положение мутации мутантного белка F RSV указано номером аминокислоты эталонной последовательности, представленной SEQ ID NO: 1.
[0021]
Лейцин, соответствующий лейцину в положении 141 аминокислотной последовательности SEQ ID NO: 1, лейцин, соответствующий лейцину в положении 142, и лейцин, соответствующий лейцину в положении 373, относятся к лейцину, соответствующему лейцину в положении 141, лейцину, соответствующему лейцину в положении 142, и лейцину, соответствующему лейцину в положении 373, в аминокислотной последовательности SEQ ID NO: 1, независимо от происхождения мутантного белка F RSV. То есть, для аминокислотной последовательности белка F RSV, в которую должна быть введена мутация, если выполняется выравнивание с аминокислотной последовательностью SEQ ID NO: 1, эти аминокислоты относятся к лейциновым остаткам, соответственно расположенным в положении лейцина 141, в положении лейцина 142 и в положении лейцина 373 аминокислотной последовательности SEQ ID NO: 1.
Так, например, в случае аминокислотной последовательности мутантного белка F RSV, N-концевая сторона которого на одну аминокислоту короче по сравнению с аминокислотной последовательностью SEQ ID NO: 1, лейцин в положении 141 в аминокислотной последовательности SEQ ID NO: 1 находится в положении 140, и такой лейцин может называться лейцином, соответствующим лейцину в положении 141 в аминокислотной последовательности SEQ ID NO: 1.
Для аминокислотной последовательности белка F RSV, в который должна быть введена мутация, при выравнивании с аминокислотной последовательностью SEQ ID NO: 1, в случае, когда аминокислота, не являющаяся лейцином, присутствует в положении лейцина, соответствующем лейцину в положении 141, в положении лейцина, соответствующем лейцину в положении 142, и/или лейцину в положении 373 аминокислотной последовательности SEQ ID NO: 1, то в мутантный белок F RSV согласно изобретению также включен вариант, в котором аминокислота заменена цистеином. Кроме того, в случае, когда аминокислота, не являющаяся лейцином, представляет собой цистеин, то замена цистеином не требуется, и аминокислота может быть цистеином сама по себе.
[0022]
Одним из вариантов осуществления изобретения является мутантный белок F RSV, который включает полипептид F2 и полипептид F1 (внеклеточную область), полученный из мутантного полипептида F0, который включает аминокислотную последовательность, где лейцин в положении 141 или лейцин в положении 142, и лейцин в положении 373 заменены цистеинами в аминокислотной последовательности SEQ ID NO: 1, и где между цистеинами образуется дисульфидная связь. В частности, в этот вариант включена аминокислотная последовательность, где в аминокислотной последовательности SEQ ID NO: 2, лейцин в положении 89 или лейцин в положении 90 и лейцин в положении 321 заменены цистеинами.
[0023]
Одним из вариантов осуществления изобретения является мутантный белок F RSV, обладающий способностью индуцировать антитело против пре-белка типа F RSV, и содержащий полипептид F2 и полипептид внеклеточной области F1, которые продуцируются из мутантного полипептида F0, имеющего аминокислотную последовательность, которая на 85% или более, предпочтительно на 90% или более, а более предпочтительно на 95% или более идентична аминокислотной последовательностью SEQ ID NO: 1, где лейцин, соответствующий лейцину в положении 141, или лейцин, соответствующий лейцину в положении 142, и лейцин, соответствующий лейцину в положении 373 аминокислотной последовательности SEQ ID NO: 1, заменены цистеинами, и где между цистеинами образуется дисульфидная связь. В частности, в этот вариант включена аминокислотная последовательность, которая на 85% или более, предпочтительно на 90% или более и более предпочтительно на 95% или более идентична аминокислотной последовательностью SEQ ID NO: 2, где лейцин, соответствующий лейцину в положении 141 (в положении 89 в SEQ ID NO: 2), или лейцин, соответствующий лейцину в положении 142 (в положении 90 в SEQ ID NO: 2), и лейцин, соответствующий лейцину в положении 373 (в положении 321 в SEQ ID NO: 2) аминокислотной последовательности SEQ ID NO: 1, заменены цистеинами.
[0024]
В настоящем описании, «процент (%) идентичности» в отношении аминокислотной последовательности определяется как процент аминокислотных остатков в последовательности-кандидате, которые идентичны аминокислотным остаткам конкретной эталонной полипептидной последовательности после выравнивания этих последовательностей, введения пробелов, если это необходимо для получения максимального % идентичности, и при этом, любая консервативная замена не рассматривается при определении идентичности последовательностей. Выравнивание для оценки % идентичности может быть достигнуто с применением различных способов, известных специалистам в данной области, например, с помощью общедоступного компьютерного программного обеспечения, такого как программное обеспечение BLAST, BLAST-2, ALIGN или Megalign (DNASTAR). Специалист в данной области может определить подходящие параметры для выравнивания последовательностей, включая любой алгоритм, необходимый для достижения максимального выравнивания по всей длине сравниваемых последовательностей. Однако, в данном случае, значение % идентичности получают с использованием компьютерной программы сравнения последовательностей BLAST при попарном выравнивании. В случае, если BLAST используется для сравнения аминокислотных последовательностей, то % идентичности данной аминокислотной последовательности A с данной аминокислотной последовательностью B вычисляют по формуле:
100 • X/Y
где X представляет собой количество аминокислотных остатков с оценками, соответствующими идентичности, как было определено с помощью выравнивания A и B в соответствии с программой BLAST для выравнивания последовательностей, а Y представляет собой общее количество аминокислотных остатков B. Если длина аминокислотной последовательности А отличается от длины аминокислотной последовательности В, то подразумевается, что % идентичности А по отношению к В отличается от % идентичности В по отношению к А. Если это не оговорено особо, то в настоящей заявке, все значения % идентичности получают с использованием компьютерной программы BLAST, как описано в предыдущем абзаце.
[0025]
Аминокислотная последовательность, имеющая предварительно определенную идентичность с заранее определенной аминокислотной последовательностью, является результатом замены, делеции, инсерции или добавления аминокислот в заранее определенную аминокислотную последовательность. Замена предпочтительно представляет собой консервативную замену. «Консервативная замена» заключается в том, что аминокислотный остаток заменяют другим химически сходным аминокислотным остатком, так, чтобы активность белка по существу не изменялась. Примеры таких замен включают случай, когда гидрофобный остаток заменен другим гидрофобным остатком; и случай, когда полярный остаток заменен другим полярным остатком, имеющим заряд, и т.п. В качестве примеров функционально сходных аминокислот, подходящих для таких замен, могут служить неполярные (гидрофобные) аминокислоты, которыми являются аланин, валин, изолейцин, лейцин, пролин, триптофан, фенилаланин, метионин и т.п. Примерами полярных (нейтральных) аминокислот являются глицин, серин, треонин, тирозин, глутамин, аспарагин, цистеин и т.п. Примерами положительно заряженных (основных) аминокислот являются аргинин, гистидин, лизин и т.п. Кроме того, примерами отрицательно заряженных (кислотных) аминокислот являются аспарагиновая кислота, глутаминовая кислота и т.п.
[0026]
Мутация глутаминовой кислоты в положении 60
Другой вариант мутантного белка F RSV согласно изобретению представляет собой мутантный белок F RSV, имеющий мутацию, где мутация представляет собой замену глутаминовой кислоты, соответствующей глутаминовой кислоте в положении 60 аминокислотной последовательности SEQ ID NO: 1, не-кислотной аминокислотой.
[0027]
Не-кислотная аминокислота не имеет конкретных ограничений при условии, что мутантный белок F RSV будет достигать эффекта предпочтительного индуцирования группы антител, которые, как считается, в значительной степени способствуют улучшению стабильности эпитопа Φ при хранении, стимулированию тримеризации и нейтрализации вируса. Однако, в частности, примерами таких аминокислот являются нейтральные аминокислоты, основные аминокислоты и неполярные (гидрофобные) аминокислоты. Более конкретно, примерами нейтральных аминокислот являются глицин, серин, треонин, тирозин, глутамин, аспарагин и цистеин; примерами основных аминокислот являются аргинин, гистидин и лизин; а примерами неполярных (гидрофобных) аминокислот являются аланин, валин, изолейцин, лейцин, пролин, триптофан, фенилаланин и метионин.
Не-кислотная аминокислота предпочтительно представляет собой нейтральную аминокислоту или неполярную (гидрофобную) аминокислоту.
Кроме того, среди нейтральных аминокислот предпочтительными являются треонин и серин, а среди неполярных (гидрофобных) аминокислот предпочтительными являются аминокислоты, выбранные из группы, состоящей из метионина, фенилаланина и лейцина. Следовательно, не-кислотную аминокислоту предпочтительно выбирают из группы, состоящей из метионина, фенилаланина, лейцина, треонина и серина.
[0028]
Одним из вариантов осуществления изобретения является мутантный белок F RSV, который включает полипептид F2 и полипептид F1 (внеклеточную область), полученный из мутантного полипептида F0, который включает аминокислотную последовательность, где глутаминовая кислота в положении 60 аминокислотной последовательности SEQ ID NO: 1 заменена не-кислотной аминокислотой. В частности, включена аминокислотная последовательность, где в аминокислотной последовательности SEQ ID NO: 2, глутаминовая кислота в положении 35 заменена не-кислотной аминокислотой.
[0029]
Одним из вариантов осуществления настоящего изобретения является мутантный белок F RSV, обладающий способностью индуцировать антитело против пре-белка типа F RSV, и содержащий полипептид F2 и полипептид F1 (внеклеточную область), которые продуцируются из мутантного полипептида F0, имеющего аминокислотную последовательность, где в аминокислотной последовательности, которая на 85% или более, предпочтительно на 90% или более, а более предпочтительно на 95% или более идентична аминокислотной последовательностью SEQ ID NO: 1, глутаминовая кислота, соответствующая глутаминовой кислоте в положении 60 аминокислотной последовательности SEQ ID NO: 1, заменена не-кислотной аминокислотой. В частности, включена аминокислотная последовательность, где в аминокислотной последовательности, которая на 85% или более, предпочтительно на 90% или более, а более предпочтительно на 95% или более идентична аминокислотной последовательности SEQ ID NO: 2, глутаминовая кислота, соответствующая глутаминовой кислоте в положении 60 (в положении 35 в SEQ ID NO: 2) аминокислотной последовательности SEQ ID NO: 1, заменена не-кислотной аминокислотой.
[0030]
Мутация глутаминовой кислоты в положении 60 может быть введена вместе с описанными выше мутациями, где лейцины заменены цистеинами.
[0031]
Мутация сайта распознавания фурином
Мутантный белок F RSV согласно изобретению может представлять собой мутантный белок F RSV, где, кроме того, аминокислота, образующая сайт распознавания фурином на С-концевой стороне области pep27, заменена так, чтобы сайт распознавания фурином не распознавался фурином. При такой аминокислотной замене, мутантный белок F RSV имеет полипептид F2 и полипептид F1 (внеклеточную область), с которым связана область pep27, и, таким образом, достигается эффект стимуляции тримеризации при сохранении стабильности эпитопа Ф при хранении.
[0032]
Область pep27 представляет собой область, соответствующую положениям 110-136 аминокислотной последовательности SEQ ID NO: 1, и первый сайт распознавания на ее N-концевой стороне и второй сайт распознавания на ее С-концевой стороне расщепляются фурином в клетке. N-концевая сторона области pep27 представляет собой полипептид F2, а С-концевая сторона представляет собой полипептид F1, и в результате расщепления образуются полипептид F2 и полипептид F1, и эти полипептиды образуют дисульфидную связь между цистеиновыми остатками, которая соответственно присуща полипептидам при образовании комплекса. Следовательно, обычно, белок F RSV не включает область pep27. Однако, из-за мутации, второй сайт распознавания не расщепляется, и только первый сайт распознавания, присутствующий между С-концевой стороной полипептида F2 и областью pep27, расщепляется фурином. В результате такого расщепления образуются полипептид, включающий полипептид F1, с которым связана область pep27, и полипептид F2, и эти полипептиды образуют дисульфидную связь между остатками цистеина, которая соответственно характерна для этих полипептидов при образовании комплекса. Следовательно, данный вариант согласно изобретению может представлять собой комплекс, который включает полипептидный фрагмент, содержащий полипептид F1, с которым связана область pep27, и полипептид F2.
[0033]
Замена может представлять собой замену одной или нескольких аминокислот, которые образуют сайты распознавания фурином. В частности, аминокислоты, которые образуют сайты распознавания фурином, представляют собой одну или несколько аминокислот, выбранных из группы, состоящей из аргинина, соответствующего аргинину в положении 133, аргинина, соответствующего аргинину в положении 135, и аргинина, соответствующего аргинину в положении 136 аминокислотной последовательности SEQ ID NO: 1, и эти аминокислоты предпочтительно заменяют не-основными аминокислотами.
Аргинин, соответствующий аргинину в положении 133, аргинин, соответствующий аргинину в положении 135, и аргинин, соответствующий аргинину в положении 136 аминокислотной последовательности SEQ ID NO: 1, могут быть независимо заменены не-основной аминокислотой.
[0034]
Не-основная аминокислота не имеет конкретных ограничений при условии, что эффект, заключающийся в стимуляции тримеризации, будет сохраняться при сохранении стабильности эпитопа Φ при хранении. Однако, в частности, примерами таких аминокислот являются нейтральные аминокислоты, кислотные аминокислоты и неполярные (гидрофобные) аминокислоты. Более конкретно, примерами нейтральных аминокислот являются глицин, серин, треонин, тирозин, глутамин, аспарагин и цистеин; примерами кислотных аминокислот являются аспарагиновая кислота и глутаминовая кислота; и примерами неполярных (гидрофобных) аминокислот являются аланин, валин, изолейцин, лейцин, пролин, триптофан, фенилаланин и метионин. Среди них, предпочтительными являются нейтральные аминокислоты, а более предпочтительным является аспарагин.
[0035]
Один вариант мутантного белка F RSV согласно изобретению включает аминокислотную последовательность, где в аминокислотной последовательности SEQ ID NO: 3, лейцин в положении 116 или лейцин в положении 117 и лейцин в положении 348 заменены цистеинами, а аминокислота, выбранная из группы, состоящей из аргинина в положении 108, аргинина в положении 110 и аргинина в положении 111, заменена неосновной аминокислотой.
[0036]
Один вариант мутантного белка F RSV согласно изобретению включает аминокислотную последовательность, где в аминокислотной последовательности, которая на 85% или более, предпочтительно на 90% или более, а более предпочтительно на 95% или более идентична аминокислотной последовательности SEQ ID NO: 3, лейцин, соответствующий лейцину в положении 141 (лейцин в положении 116 в SEQ ID NO: 3), или лейцин, соответствующий лейцину в положении 142 (лейцин в положении 117 в SEQ ID NO: 3), и лейцин, соответствующий лейцину в положении 373 (лейцин в положении 348 в SEQ ID NO: 3) аминокислотной последовательности SEQ ID NO: 1, заменены цистеинами, и аминокислота, выбранная из группы, состоящей из аргинина, соответствующего аргинину в положении 133 (аргинину в положении 108 в SEQ ID NO: 3), аргинина, соответствующего аргинину в положении 135 (аргинину в положении 110 в SEQ ID NO: 3), и аргинина, соответствующего аргинину в положении 136 (аргинину в положение 111 в SEQ ID NO: 3) аминокислотной последовательности SEQ ID NO: 1, заменена не-основной аминокислотой.
[0037]
Другой вариант мутантного белка F RSV согласно изобретению включает аминокислотную последовательность, где в аминокислотной последовательности SEQ ID NO: 3, глутаминовая кислота в положении 35 заменена не-кислотной аминокислотой, и аминокислота, выбранная из группы, состоящей из аргинина в положении 108, аргинина в положении 110 и аргинина в положении 111, заменена не-основной аминокислотой.
[0038]
Другой вариант мутантного белка F RSV согласно изобретению включает аминокислотную последовательность, где в аминокислотной последовательности, которая на 85% или более, предпочтительно на 90% или более, а более предпочтительно на 95% или более идентична аминокислотной последовательности SEQ ID NO: 3, глутаминовая кислота, соответствующая глутаминовой кислоте в положении 60 в аминокислотной последовательности SEQ ID NO: 1 (глутаминовой кислоте в положении 35 в SEQ ID NO: 3) заменена не-кислотной аминокислотой, а аминокислота, выбранная из группы, состоящей из аргинина, соответствующего аргинину в положении 133 (аргинину в положении 108 в SEQ ID NO: 3), аргинина, соответствующего аргинину в положении 135 (аргинину в положении 110 в SEQ ID NO: 3), и аргинина, соответствующего аргинину в положении 136 (аргинину в положении 111 в SEQ ID NO: 3) аминокислотной последовательности SEQ ID NO: 1, заменена не-основной аминокислотой.
[0039]
В SEQ ID NO: 3 или в аминокислотной последовательности, которая на 85% или более, предпочтительно на 90% или более, и более предпочтительно на 95% или более идентична SEQ ID NO: 3, область pep27 может быть заменена искусственным линкером. Последовательность искусственного линкера не имеет конкретных ограничений, но ее длина предпочтительно составляет 6-27 аминокислотных остатков, при этом, предпочтительной является последовательность, содержащая Gly и/или Ser. Более конкретно, их примеры включают Gly-Ser-Gly-Ser-Gly-Ser (SEQ ID NO: 18), (Gly-Gly-Gly-Gly-Ser)2 (SEQ ID NO: 19) и (Gly-Gly-Gly-Gly-Ser)3 (SEQ ID NO: 20).
[0040]
Другие мутации
Мутантный белок F RSV согласно изобретению может представлять собой мутантный белок F RSV, где, в дополнение к мутации, в которой лейцин, соответствующий лейцину в положении 141, или лейцин, соответствующий лейцину в положении 142, и лейцин, соответствующий лейцину в положении 373 аминокислотной последовательности SEQ ID NO: 1 заменен цистеинами (далее эта мутация будет называться мутацией, при которой лейцины заменены цистеинами), имеется мутация глутаминовой кислоты в положении 60 и/или мутация сайта распознавания фурином, описанная выше, и кроме того, аминокислота, соответствующая треонину в положении 189, и/или аминокислота, соответствующая серину в положении 190 аминокислотной последовательности SEQ ID NO: 1, заменены гидрофобными аминокислотами.
Мутантный белок F RSV согласно варианту осуществления изобретения дает эффект, заключающийся в том, что уровень экспрессии в экспрессионной системе значительно повышается, при этом, также сохраняется эффект превосходной стабильности эпитопа Φ при хранении и стимуляции тримеризации, а также эффект предпочтительного индуцирования группы антител, которые, как считается, обеспечивают высокую степень нейтрализации вируса.
Аминокислота, соответствующая треонину в положении 189, и аминокислота, соответствующая серину в положении 190 аминокислотной последовательности SEQ ID NO: 1, могут быть независимо заменены гидрофобной аминокислотой.
Кроме того, мутантный белок F RSV, содержащий замену, трудно распознается антителом, которое неэффективно или плохо нейтрализует вирусную инфекцию, и кроме того, дает эффект значительного повышения уровня экспрессии в экспрессионной системе, сохранения превосходной стабильности эпитопа Φ при хранении и стимуляции тримеризации, а также эффект предпочтительного индуцирования группы антител, которые, как считается, обеспечивают высокую степень нейтрализацию вируса. И наоборот, если индуцируется антитело, которое неэффективно или плохо нейтрализует вирусную инфекцию, то существует ожидаемый риск того, что иммунная клетка, которая не способствует или почти не способствует нейтрализации вирусной инфекции, будет активироваться, что будет приводить к обострению симптомов. Однако, как видно из примеров, описанных ниже, мутантный белок F RSV в соответствии с вариантом осуществления изобретения, трудно распознается антителом, которое неэффективно или плохо нейтрализует вирусную инфекцию, и, таким образом, риск в случае мутантного белка F RSV согласно изобретению будет ниже, чем в случае применения обычной вакцины.
В качестве конкретного примера, мутантный белок F RSV согласно варианту осуществления изобретения с трудом распознается антителом, которое распознает эпитоп I. Поскольку эпитоп I индуцирует антитело, обладающее очень низкой способностью нейтрализовать вирусную инфекцию, то ожидается, что риск того, что эпитоп I будет распознаваться антителом, и иммунная клетка почти не будет нейтрализовать вирусную инфекцию, будет повышаться, что таким образом, будет приводить к обострению симптомов. Однако, как видно из примеров, описанных ниже, в случае использования мутантного белка F RSV согласно варианту осуществления изобретения, который трудно распознается антителом, распознающим эпитоп I, риск будет ниже, чем в случае обычной вакцины.
[0041]
Гидрофобная аминокислота не имеет конкретных ограничений при условии, что в дополнение к эффекту значительного повышения уровня экспрессии в экспрессионной системе, сохранения превосходной стабильности эпитопа Φ при хранении и стимуляции тримеризации, а также к эффекту предпочтительного индуцирования группы антител, которые, как считается, обеспечивают высокую степень нейтрализацию вируса, этот мутантный белок F RSV будет с трудом распознаваться антителом, которое неэффективно или плохо нейтрализует вирусную инфекцию. Однако, в частности, примерами таких аминокислот являются аланин, валин, изолейцин, лейцин, пролин, триптофан, фенилаланин и метионин. Среди них, предпочтительными являются аминокислоты, выбранные из группы, состоящей из валина, изолейцина и лейцина.
[0042]
Положение 189 в SEQ ID NO: 1 соответствует аминокислоте в положении 137 в SEQ ID NO: 2, а положение 190 в SEQ ID NO: 1 соответствует аминокислоте в положении 138 в SEQ ID NO: 2. В SEQ ID NO: 2, или в аминокислотной последовательности, которая на 85% или более, на 90% или более или на 95% или более идентична SEQ ID NO: 2, мутация одной или обеих этих аминокислот может быть введена в дополнение к мутации, при которой лейцины заменены цистеинами и/или мутации глутаминовой кислоты в положении 60, описанной выше.
Кроме того, положение 189 в SEQ ID NO: 1 соответствует аминокислоте в положении 164 в SEQ ID NO: 3, а положение 190 в SEQ ID NO: 1 соответствует аминокислоте в положении 165 в SEQ ID NO: 3. В SEQ ID NO: 3, или в аминокислотной последовательности, которая на 85% или более, на 90% или более или на 95% или более идентична SEQ ID NO: 3, мутация одной или обеих этих аминокислот может быть введена в дополнение к мутации, при которой лейцины заменены цистеинами, мутации глутаминовой кислоты в положении 60, и/или мутации сайта распознавания фурином, как описано выше.
[0043]
Одним из вариантов настоящего изобретения может быть мутантный белок F RSV, в котором, в дополнение к мутации, при которой лейцины заменены цистеинами, присутствует мутация глутаминовой кислоты в положении 60 и/или мутация сайта распознавания фурином, как описано выше, и в котором, в аминокислотной последовательности SEQ ID NO: 1, аминокислота, соответствующая лизину в положении 42, заменена на аргинин, и/или аминокислота, соответствующая валину в положении 384, заменена на треонин.
Мутантный белок F RSV согласно варианту осуществления изобретения обеспечивает эффект, заключающийся в том, что уровень экспрессии в экспрессионной системе значительно повышается, при этом, также сохраняется эффект превосходной стабильности эпитопа Φ при хранении и стимуляция тримеризации, а также эффект предпочтительного индуцирования группы антител, которые, как считается, обладают высокой способностью к нейтрализации вируса.
[0044]
Положение 42 в SEQ ID NO: 1 соответствует аминокислоте в положении 17 в SEQ ID NO: 2, а положение 384 в SEQ ID NO: 1 соответствует аминокислоте в положении 332 в SEQ ID NO: 2. В SEQ ID NO: 2 или в аминокислотной последовательности, которая на 90% или более идентична SEQ ID NO: 2, мутация одной или обеих этих аминокислот может быть введена в дополнение к мутации, при которой лейцины заменены цистеинами, и/или мутации глутаминовой кислоты в положении 60, описанной выше, и в дополнение к мутации в положении 189 и/или в положении 190, как описано выше.
Кроме того, положение 42 в SEQ ID NO: 1 соответствует аминокислоте в положении 17 в SEQ ID NO: 3, а положение 384 в SEQ ID NO: 1 соответствует аминокислоте в положении 359 в SEQ ID NO: 3. В SEQ ID NO: 3 или в аминокислотной последовательности, которая на 90% или более идентична SEQ ID NO: 3, мутация одной или обеих этих аминокислот может быть введена в дополнение к мутации, при которой лейцины заменены цистеинами, мутации глутаминовой кислоты в положении 60, и/или мутации сайта распознавания фурином как описано выше, и в дополнение к мутации в положении 189 и/или в положении 190, как описано выше.
[0045]
Мутантный белок F RSV может дополнительно включать общеизвестные мутации, при условии, что эти мутации не будут влиять на эффекты мутантного белка F RSV. Так, например, дополнительно может быть включена мутация для повышения уровня экспрессии в экспрессионной системе. В частности, могут быть включены одна или несколько замен, выбранных из группы, состоящей из замены аминокислоты, соответствующей пролину в положении 102 аминокислотной последовательности SEQ ID NO: 1, на аланин, замены аминокислоты, соответствующей изолейцину в положении 379, валином, и замены аминокислоты, соответствующей метионину в положении 447, валином.
[0046]
Положение 102 в SEQ ID NO: 1 соответствует аминокислоте в положении 77 в SEQ ID NO: 2, положение 379 в SEQ ID NO: 1 соответствует аминокислоте в положении 327 в SEQ ID NO: 2, а положение 447 в SEQ ID NO: 1 соответствует аминокислоте в положении 395 в SEQ ID NO: 2. В SEQ ID NO: 2 или в аминокислотной последовательности, которая на 85%, 90% или 95% или более идентична SEQ ID NO: 2, мутация одной или обеих из этих аминокислот может быть введена в дополнение к мутации, при которой лейцины заменены цистеинами, и/или мутации глутаминовой кислоты в положении 60, описанной выше, и, кроме того, в дополнение к мутации в положении 189 и/или в положении 190, или в положении 42, и/или в положении 384, как описано выше.
Кроме того, положение 102 в SEQ ID NO: 1 соответствует аминокислоте в положении 77 в SEQ ID NO: 3, положение 379 в SEQ ID NO: 1 соответствует аминокислоте в положении 354 в SEQ ID NO: 3, а положение 447 в SEQ ID NO: 1 соответствует аминокислоте в положении 422 в SEQ ID NO: 4. В SEQ ID NO: 3 или в аминокислотной последовательности, которая на 85%, 90% или 95% или более идентична SEQ ID NO: 3, мутация одной или обеих из этих аминокислот может быть введена в дополнение к мутации, при которой лейцины заменены цистеинами, мутации глутаминовой кислоты в положении 60, и/или мутации сайта распознавания фурином, как описано выше, и, кроме того, в дополнение к мутации в положении 189 и/или или в положении 190, или в положении 42, и/или в положении 384, как описано выше.
[0047]
Конкретным вариантом мутантного белка F RSV согласно изобретению является мутантный белок F RSV, включающий аминокислотную последовательность любой из SEQ ID NO: 6-9. В частности, были включены нижеследующие аминокислотные мутации.
[0048]
Аминокислотная последовательность SEQ ID NO: 6 (мутантный белок RSV_F, содержащийся в FH_50) представляет собой аминокислотную последовательность, в которой в SEQ ID NO: 3, лейцин в положении 141 SEQ ID NO: 1 заменен цистеином; лейцин в положении 373 SEQ ID NO: 1 заменен цистеином; аргинин в положении 135 SEQ ID NO: 1 заменен аспарагином; аргинин в положении 136 SEQ ID NO: 1 заменен аспарагином; треонин в положении 189 SEQ ID NO: 1 заменен валином; серин в положении 190 SEQ ID NO: 1 заменен валином; пролин в положении 102 SEQ ID NO: 1 заменен аланином; изолейцин в положении 379 SEQ ID NO: 1 заменен валином; а метионин в положении 447 SEQ ID NO: 1 заменен валином. В данном случае, аминокислотная последовательность SEQ ID NO: 6 фактически разделена на аминокислотную последовательность полипептида F2 и аминокислотную последовательность полипептида F1 с добавлением pep27 (внеклеточной области). Однако, в данном случае, они представлены в виде одной аминокислотной последовательности, в которой они соединены вместе. Таким образом, при определении идентичности аминокислотных последовательностей, описанных ниже, проводят сравнение с аминокислотной последовательностью, в которой аминокислотная последовательность полипептида F2 и аминокислотная последовательность полипептида F1 с добавлением pep27 (внеклеточной области) белка-мишени связаны в указанном порядке. То же самое применимо к последовательностям, представленным ниже.
[0049]
Аминокислотная последовательность SEQ ID NO: 7 (мутантный белок RSV_F, содержащийся в FH_81) представляет собой аминокислотную последовательность, в которой, в SEQ ID NO: 3, лейцин в положении 141 SEQ ID NO: 1 заменен цистеином; лейцин в положении 373 SEQ ID NO: 1 заменен цистеином; аргинин в положении 135 SEQ ID NO: 1 заменен аспарагином; аргинин в положении 136 SEQ ID NO: 1 заменен аспарагином; треонин в положении 189 SEQ ID NO: 1 заменен валином; серин в положении 190 SEQ ID NO: 1 заменен валином; глутаминовая кислота в положении 60 SEQ ID NO: 1 заменена метионином; пролин в положении 102 SEQ ID NO: 1 заменен аланином; изолейцин в положении 379 SEQ ID NO: 1 заменен валином; а метионин в положении 447 SEQ ID NO: 1 заменен валином.
[0050]
Аминокислотная последовательность SEQ ID NO: 8 (мутантный белок RSV_F, содержащийся в FH_82) представляет собой аминокислотную последовательность, в которой в SEQ ID NO: 3, лейцин в положении 141 SEQ ID NO: 1 заменен цистеином; лейцин в положении 373 SEQ ID NO: 1 заменен цистеином; аргинин в положении 135 SEQ ID NO: 1 заменен аспарагином; аргинин в положении 136 SEQ ID NO: 1 заменен аспарагином; треонин в положении 189 SEQ ID NO: 1 заменен валином; серин в положении 190 SEQ ID NO: 1 заменен валином; лизин в положении 42 SEQ ID NO: 1 заменен аргинином; валин в положении 384 SEQ ID NO: 1 заменен треонином; пролин в положении 102 SEQ ID NO: 1 заменен аланином; изолейцин в положении 379 SEQ ID NO: 1 заменен валином; а метионин в положении 447 SEQ ID NO: 1 заменен валином.
[0051]
Аминокислотная последовательность SEQ ID NO: 9 (мутантный белок RSV_F, содержащийся в FH_85) представляет собой аминокислотную последовательность, в которой в SEQ ID NO: 3, лейцин в положении 141 SEQ ID NO: 1 заменен цистеином; лейцин в положении 373 SEQ ID NO: 1 заменен цистеином; аргинин в положении 135 SEQ ID NO: 1 заменен аспарагином; аргинин в положении 136 SEQ ID NO: 1 заменен аспарагином; треонин в положении 189 SEQ ID NO: 1 заменен валином; серин в положении 190 SEQ ID NO: 1 заменен валином; глутаминовая кислота в положении 60 SEQ ID NO: 1 заменена метионином; лизин в положении 42 SEQ ID NO: 1 заменен аргинином; валин в положении 384 SEQ ID NO: 1 заменен треонином; пролин в положении 102 SEQ ID NO: 1 заменен заменен аланином; изолейцин в положении 379 SEQ ID NO: 1 заменен валином; а метионин в положении 447 SEQ ID NO: 1 заменен валином.
[0052]
Один вариант осуществления изобретения представляет собой мутантный белок F RSV, обладающий способностью индуцировать антитело против пре-белка типа F RSV и содержащий описанные выше аминокислотные замены в аминокислотной последовательности, которая на 85% или более, предпочтительно на 90% или более и более предпочтительно на 95% или более идентична аминокислотной последовательности любой из SEQ ID NO: 6-9, где между цистеинами образуется дисульфидная связь.
Определение аминокислотной последовательности, которая идентична уже определенной аминокислотной последовательности, является таким же, как описано выше.
[0053]
Слитый белок
Другой вариант настоящего изобретения представляет собой слитый белок, который содержит мутантный белок F RSV и по меньшей мере домен мультимеризации.
Слитый белок может содержать полипептид, отличающийся от мутантного белка F RSV, и домен мультимеризации. Полипептид, отличающийся от мутантного белка F RSV и домен мультимеризации, не имеют конкретных ограничений, при условии, что они не будут негативно влиять на действие мутантного белка F RSV. Однако, в частности, примерами таких белков являются мотивы, такие, как мотив разрезания тромбином, и метки для очистки, такие как His-метка или Strep-метка II.
[0054]
Домен мультимеризации не имеет конкретных ограничений, при условии, что он будет представлять собой полипептид, необходимый для мультимеризации мутантного белка F RSV. Однако, в качестве примера можно привести фолдоновый домен для тримеризации (Yizhi Tao et al., 1997, Structure 5 (6): 789) (Frank S. et al., 2001, Journal of molecular biology 308 (5): 1081).
Примером фолдонового домена может служить фолдоновый домен, полученный из фибритина бактериофага Т4. В качестве его аминокислотной последовательности может служить аминокислотная последовательность SEQ ID NO: 21.
[0055]
Примером слитого белка является слитый белок, в котором фолдоновый домен связан с С-концом мутантного белка F RSV, имеющего аминокислотную последовательность SEQ ID NO: 6-9.
В частности, примерами таких белков являются белки, имеющие аминокислотные последовательности SEQ ID NO: 10-13.
Фактически, аминокислотная последовательность SEQ ID NO: 10-13 разделена на аминокислотную последовательность полипептида F2 и аминокислотную последовательность полипептида F1 с добавлением pep27 (внеклеточной области). Однако, в данном случае, они представлены в виде одной аминокислотной последовательности, в которой они соединены вместе.
[0056]
Мультимер
Другой вариант настоящего изобретения представляет собой мультимер слитого белка. В мультимере, слитый белок связан посредством домена мультимеризации, такого как домен фолдона. Мультимер предпочтительно представляет собой тример слитого белка.
[0057]
Полинуклеотид
Другой вариант настоящего изобретения представляет собой полинуклеотид, кодирующий мутантный белок F RSV или слитый белок.
Полинуклеотид, кодирующий мутантный белок F RSV, может быть получен в виде полинуклеотида, в который была введена желаемая мутация с применением стандартной методики генной инженерии исходя из полинуклеотидной последовательности (SEQ ID NO: 74), кодирующей белок F RSV до введения мутации.
Кроме того, полинуклеотид, кодирующий слитый белок, также может быть получен с применением стандартной методики генной инженерии с использованием полинуклеотида, кодирующего мутантный белок F RSV, и полинуклеотида, кодирующего полипептид, отличающийся от мутантного белка F RSV.
[0058]
В частности, примеры полинуклеотидной последовательности, которую необходимо использовать для экспрессии слитого белка согласно изобретению и его продуцирования у хозяина, включают полинуклеотидные последовательности SEQ ID NO: 14-17.
SEQ ID NO: 14 представляет собой полинуклеотидную последовательность, предназначенную для экспрессии слитого белка, имеющего аминокислотную последовательность SEQ ID NO: 10, и его продуцирования у хозяина. Однако, к 5'-концу последовательности, кодирующей слитый белок, присоединяют последовательность, кодирующую сигнальный пептид.
SEQ ID NO: 15 представляет собой полинуклеотидную последовательность, предназначенную для экспрессии слитого белка, имеющего аминокислотную последовательность SEQ ID NO: 11, и его продуцирования у хозяина. Однако, к 5'-концу последовательности, кодирующей слитый белок, присоединяют последовательность, кодирующую сигнальный пептид.
SEQ ID NO: 16 представляет собой полинуклеотидную последовательность, предназначенную для экспрессии слитого белка, имеющего аминокислотную последовательность SEQ ID NO: 12, и его продуцирования у хозяина. Однако, к 5'-концу последовательности, кодирующей слитый белок, присоединяют последовательность, кодирующую сигнальный пептид.
SEQ ID NO: 17 представляет собой полинуклеотидную последовательность, предназначенную для экспрессии слитого белка, имеющего аминокислотную последовательность SEQ ID NO: 13, и его продуцирования у хозяина. Однако, к 5'-концу последовательности, кодирующей слитый белок, присоединяют последовательность, кодирующую сигнальный пептид.
[0059]
Может быть использован любой кодон, при условии, что он будет кодировать предполагаемую аминокислоту. Так, например, существует четыре типа кодонов, кодирующих Gly: GGT, GGC, GGA и GGG, и может быть использован любой из них. Кроме того, каждый кодон может быть оптимизирован или не оптимизирован, но предпочтительно, чтобы был оптимизирован каждый кодон.
[0060]
Полинуклеотид, кодирующий мутантный белок F RSV, может представлять собой полинуклеотид, который гибридизуется в жестких условиях с полинуклеотидом, имеющим последовательность, комплементарную этим полинуклеотидным последовательностям, при условии, что полинуклеотид, кодирующий мутантный белок F RSV, будет кодировать мутантный белок F RSV, который включает вышеуказанные специфические мутации и обладает способностью индуцировать антитело против пре-белка типа F RSV. В данном случае, примером жесткого условия является условие проведения промывки при концентрации соли, соответствующей 68°C, 0,1×SSC, 0,1% ДСН после Саузерн-гибридизации.
[0061]
Экспрессионный кластер
Другой вариант настоящего изобретения представляет собой экспрессионный кластер, включающий полинуклеотид.
Экспрессионный кластер может экспрессировать полинуклеотид по определенному механизму, например, он может включать элемент, необходимый для экспрессии промоторной последовательности, последовательности сигнала терминации транскрипции и т.п., и может представлять собой экспрессионный кластер, используемый в стандартной методике генной инженерии. Экспрессионный кластер может быть включен, например, в рекомбинантный вектор. Примерами рекомбинантного вектора являются плазмидный вектор и вирусный вектор, и примерами таких векторов являются вектор, который может экспрессироваться в прокариотической клетке; вектор, который может экспрессироваться в эукариотической клетке; и вектор, который может экспрессироваться в клетке, происходящей от млекопитающего.
[0062]
Клетка-хозяин
Другой вариант настоящего изобретения представляет собой клетку-хозяина, которая включает экспрессионный кластер. Предпочтительно, клетка-хозяин представляет собой клетку-хозяина, трансформированную рекомбинантным вектором, включающим экспрессионный кластер.
Клетка-хозяин не имеет конкретных ограничений, при условии, что белок, кодируемый полинуклеотидом, будет экспрессироваться из экспрессионного кластера, и что ее можно будет использовать в стандартной методике генной инженерии. В частности, примерами таких клеток являются прокариотические клетки, такие как Escherichia coli и Bacillus subtilis, эукариотические клетки и клетки, происходящие от млекопитающих.
Введение полинуклеотида в клетку-хозяина может быть осуществлено с применением общеизвестных методов, таких как метод с использованием фосфата кальция, метод с использованием DEAE-декстрана, метод электропорации и метод липофекции.
При культивировании клетки-хозяина в подходящих условиях может быть экспрессирован мутантный белок F RSV или слитый белок, и может быть получен мутантный белок F RSV, слитый белок или их мультимер. Полученный мутантный белок F RSV, слитый белок или их мультимер могут быть очищены общеизвестными способами.
[0063]
Иммуноген
Другой вариант настоящего изобретения представляет собой иммуноген, который включает мутантный белок F RSV, слитый белок, мультимер или описанный ниже продукт в виде частиц.
То есть, мутантный белок F RSV, слитый белок, мультимер или продукт в виде частиц могут быть использованы в качестве иммуногена. Иммуноген может включать любой один, любые два или все из нижеперечисленных белков, таких, как мутантный белок F RSV, слитый белок, мультимер или продукт в виде частиц, при условии, что иммуноген не будет негативно влиять на действие мутантного белка F RSV. Среди них, предпочтительным является мультимер, а особенно тример, а продукт в виде частиц является предпочтительным для его использования в качестве иммуногена. Получение мультимера описано выше, а продукт в виде частиц будет подробно описан ниже. Иммуноген согласно изобретению может содержать консервант, наполнитель и т.п., входящие в состав стандартного иммуногена.
[0064]
Продукт в виде частиц
Другой вариант настоящего изобретения представляет собой продукт в виде частиц мутантного белка F RSV или мультимера.
Продукт в виде частиц не имеет конкретных ограничений, при условии, что он будет представлять собой частицы, образованные посредством агрегации двух или более мутантных белков или мультимеров F RSV, и может эффективно индуцировать антитело, обладающее высокой нейтрализующей способностью. Однако, например, продукт в виде частиц представляет собой частицу, образованную посредством агрегации двух или более мутантных белков F RSV или мультимеров посредством домена, образующего частицы. В данном случае, формы продукта в виде частиц и самих частиц не обязательно должны ограничиваться сферическими формами, и достаточно, чтобы их агрегация происходила лишь в определенном порядке.
То есть, предпочтительно, чтобы домен, образующий частицы, был также слвязан с мутантным белком F RSV или слитым белком, а мультимер мутантного белка F RSV или слитого белка был агрегирован посредством образующего частицы домена с образованием частиц.
Предпочтительно, чтобы образование частиц осуществлялось с использованием слитого белка для получения продукта в виде частиц, в котором образующий частицы домен связан с С-концом мутантного белка F RSV или слитого белка. Слитый белок для получения продукта в виде частиц будет описан ниже.
[0065]
В продукте, представляющем собой частицы, образующий частицы домен предпочтительно расположен ближе к эпитопу I, чем к эпитопу Φ, в стерической структуре мутантного белка F RSV или мультимера. То есть, предпочтительным продуктом является продукт в виде частиц, в котором ядро образуется посредством агрегации образующего частицы домена, а эпитоп I мутантного белка F RSV или мультимера присутствуют на внутренней стороне, и эпитоп Φ подвергается воздействию с внешней стороны.
Таким образом, продукт в виде частиц согласно одному из вариантов изобретения представляет собой продукт в виде частиц, в котором два или более мутантных белков F RSV или мультимеров агрегируются посредством домена, образующего частицы, в результате чего по меньшей мере эпитоп Φ белка F RSV подвергается воздействию и по меньшей мере эпитоп I белка F RSV не подвергается такому воздействию. Используемый здесь термин «эпитоп I не подвергается воздействию» необязательно означает, что эпитоп I полностью не подвергается воздействию до тех пор, пока не будет снижаться доступность антител или различных иммунных клеток к эпитопу I.
[0066]
В одном варианте продукта в виде частиц согласно изобретению, поскольку эпитоп Φ и эпитоп I мутантного белка F RSV расположены на двух крайних точках стерической структуры белка, то образующий частицу домен, который образует сердцевину, расположен ближе к эпитопу I, чем к эпитопу Φ, и, таким образом, может быть получена структура, в которой эпитоп I, являющийся ненужным эпитопом, замаскирован под антиген, а эпитоп Φ, являющийся полезным эпитопом, может легко функционировать в качестве антигена. В данном случае, ненужный эпитоп означает эпитоп, который обычно индуцирует антитело, обладающее низкой способностью нейтрализовать вирусную инфекцию RSV у человека, если мутантный белок F RSV, его слитый белок, мультимер или продукт в виде частиц используют в качестве иммуногена. Конкретными примерами является эпитоп I. С другой стороны, полезный эпитоп означает эпитоп, который обычно индуцирует антитело, обладающее высокой способностью нейтрализовать вирусную инфекцию RSV у человека, если мутантный белок F RSV, его слитый белок, мультимер или продукт в виде частиц используют в качестве иммуногена. Конкретные примеры таких эпитопов включают эпитоп Φ и эпитоп V. В результате образования такой структуры достигается эффект, заключающийся в том, что доступ антител или различных иммунных клеток к ненужному эпитопу снижается за счет эффекта стерических затруднений.
[0067]
Домен, образующий частицы, представляет собой полипептид, который превращает мутантный белок F RSV или его мультимер в частицы. Примерами являются домены, которые могут связываться с каркасными частицами (например, пептиды, связывающиеся с каркасными частицами), домены, которые могут гидрофобно агрегироваться (например, гидрофобные пептидные цепи), домены, которые обладают способностью к самоассоциации (например, самоассоциирующиеся пептиды) или т.п.
[0068]
В данном разделе, в первую очередь приводится описание варианта образования частиц с использованием каркасных частиц. То есть, этот вариант представляет собой мутантный белок F RSV или мультимер, связанный с каркасными частицами.
Состав каркасных частиц не имеет конкретных ограничений. Однако, их примерами являются белки, обладающие способностью образовывать частицы, липидные бислои, оболочки, липосомы, ниосомы, виросомы, ферросомы, золотые наночастицы, смолы, диоксид кремния, полимерные мицеллы, гели и т.п.
Примерами белков, обладающих способностью образовывать частицы, являются вирусные белки или варианты вирусных белков, которые образуют VLP (вирусоподобные частицы) или VP (вирусные частицы) (см., например, выложенную публикацию Японской патентной заявки № 2004-2313). Так, например, можно упомянуть белок поверхностного антигена вируса гепатита В, модифицированный для удаления исходной инфекционной способности по отношению к гепатоцитам и дополнительно модифицированный для представления каркасной молекулы, а при экспрессии в эукариотических клетках, этот белок экспрессируется и накапливается в виде белка на мембране, такой как мембрана эндоплазматического ретикулума, и высвобождается в виде VLP.
Более конкретно, в качестве каркасных частиц можно упомянуть VLP белкового происхождения, полученные путем встраивания Fc-связывающего домена (Z-домена) белка A в PreS-область поверхностного антигена вируса гепатита B (антигена HBs), и, например, может быть использована бионанокапсула-ZZ (номер по каталогу Beacle: BCL-DC-002).
Предпочтительно, чтобы молекула (каркасная молекула), которая связывается с пептидом, связывающимся с каркасной частицей, в качестве домена, образующего частицу, была иммобилизована на поверхности каркасной частицы.
Так, например, можно упомянуть Fc-связывающий домен белка А, а мутантный белок F RSV или его мультимер может быть связан посредством пептида, связывающегося с каркасными частицами.
[0069]
Связь между каркасной молекулой и пептидом, связывающимся с каркасной частицей, может быть либо ковалентной, либо нековалентной. Примеры получения ковалентных связей включают методы, в совокупности называемые «клик-химией» (примерами являются алкиновая (например, ацетилен)-азидная связь, цистеин-малеимидная связь и связь первичного амина с NHS-эфиром), метод образования связи со сдвигом заряда (примером является гидразид-альдегидная связь), способ создания ковалентной связи в зависимости от полипептида (такой как способ с использованием SpyTag-SpyCatcher) и т.п. Примерами нековалентных связей являются ионное взаимодействие, гидрофобная связь, водородная связь и т.п.
В частности, примерами являются комбинации белковых меток и молекул, связывающих белковые метки (биотин-авидин, Fc-белок А, GST (глутатион-S-трансфераза)-глутатион, MBP (белок, связывающийся с мальтозой)-мальтоза, His-метка-никель и метка SBP-стрептавидин), комбинация антитела и антигена и т.п.
Предпочтительная комбинация каркасной молекулы и пептида, связывающегося с каркасной частицей, представляет собой комбинацию Fc-домена IgG1 (SEQ ID NO: 30) и Fc-связывающего домена (Z-домена) белка А. Fc-домен IgG1 может быть использован для каркасной молекулы, а Fc-связывающий домен белка А может быть использован для пептида, связывающего каркасную частицу. Fc-домен IgG1 может быть использован в качестве пептида, связывающего каркасную частицу, а Fc-связывающий домен белка А может быть использован в качестве каркасной молекулы.
Поскольку вышеуказанные каркасные молекулы или пептиды, связывающиеся с каркасными частицами, могут быть ковалентно или нековалентно связаны друг с другом, то могут быть использованы их неполные фрагменты, либо они могут быть модифицированы.
[0070]
Далее описывается вариант образования частиц с использованием гидрофобной пептидной цепи в качестве домена, образующего частицы.
В этом варианте, гидрофобная пептидная цепь связана с мутантным белком F RSV или слитым белком, и мутантный белок F RSV или слитый белок образует частицу посредством гидрофобной пептидной цепи.
Гидрофобная пептидная цепь означает пептидную цепь, которая содержит гидрофобную аминокислоту и подвергается самосборке с образованием продукта в виде частиц, и которая содержит 5-50, а предпочтительно 10-30 аминокислотных остатков. В данном случае, примерами в высокой степени гидрофобных аминокислот являются валин, лейцин, фенилаланин, изолейцин и т.п., а более предпочтительными являются лейцин или изолейцин. Соотношение гидрофобных аминокислот в гидрофобной пептидной цепи предпочтительно составляет 20-60%.
В гидрофобной пептидной цепи предпочтительно, чтобы гидрофобные аминокислоты находились на поверхности гидрофобной пептидной цепи. Доля гидрофобных аминокислот на поверхности предпочтительно составляет 10% или более. Однако, если соотношение слишком велико, то эти аминокислоты агрегируются внутри клеток и не секретируется, и, таким образом, гидрофобные аминокислоты на поверхности предпочтительно составляют 50% или менее, 30% или менее и 20% или менее. С другой стороны, более высокое соотношение гидрофобных аминокислот на внутренней стороне приводит к образованию стабильной структуры гидрофобной пептидной цепи и, таким образом, является предпочтительным.
При этом, может быть использована любая аминокислота, кроме гидрофобной аминокислоты. Однако, в частности, для усиления секреторного эффекта предпочтительно, чтобы содержание гистидина составляло 20% или более.
При формирования гидрофобной пептидной цепи в виде повторяющейся последовательности пептида, состоящей из 7 аминокислот, образуется спиральная структура, и описанная выше конструкция может быть легко получена, а поэтому предпочтительно использовать именно ее. Так, например, гидрофобная пептидная цепь предпочтительно представляет собой повторяющуюся пептидную последовательность, состоящую из 7 аминокислот, в которой гидрофобные аминокислоты расположены в положениях 1, 3 и 5. Количество повторов составляет, например, 2-5, а предпочтительно 2-3.
Предпочтительные примеры гидрофобной пептидной цепи включают CC02, CC03, CC07 и CC08 (в указанном порядке, SEQ ID NO: 40, 41, 42 и 43), и среди них особенно предпочтительной является CC07 (SEQ ID NO: 42).
[0071]
Далее описывается вариант образования частиц с использованием домена самоассоциирующегося белка в качестве домена, образующего частицы.
В этом варианте осуществления изобретения, домен самоассоциирующегося белка связан с мутантным белком F RSV или слитым белком, и этот мутантный белок F RSV или слитый белок образует частицу посредством домена самоассоциирующегося белка.
Примерами самоассоциирующихся белков являются коровый антиген вируса гепатита В (HBcAg), капсидный белок вируса папилломы человека (HPV L1), капсидный белок вируса гепатита Е (капсид HEV), белок оболочки Qβ бактериофага, капсид вируса норуолк (норовируса, NoV), капсид парвовируса В19, вирус мозаики люцерны, феритин, инкапсулин и т.п. Самоассоциирующийся белок может представлять собой самоассоциирующийся частичный фрагмент или вариант.
В случае, если используется HBcAg, то при связывании с белком F RSV или с мультимером согласно изобретению предпочтительно, чтобы белок F RSV или его мультимер связывался с сайтом, удаленным от поверхности ассоциации для самоассоциации HbcAg, и на внешней стороне самоассоциированных белков. Кроме того, при осуществлении такого связывания, предпочтительно встраивать линкер между спиралью домена α3 и спиралью домена α4, чтобы получить исходную стерическую структуру HBcAg. Линкер представляет собой, например, полипептид, содержащий 10-30 аминокислот, и, например, линкер GS. Примером последовательности, в которой метка FLAG связана с таким вариантом HBcAg, является SEQ ID NO: 54.
[0072]
<Слитый белок для получения продукта в виде частиц>
Слитый белок для получения продукта в виде частиц представляет собой белок, используемый для получения указанного выше продукта в виде частиц, и содержит домен, образующий частицы, такой как каркасный пептид, связывающийся с частицами, гидрофобную пептидную цепь или домен самоассоциирующегося белка, описанный выше, который связан с С-концом мутантного белка F RSV согласно изобретению или с С-концом слитого белка.
Слитый белок согласно изобретению, используемый для получения продукта в виде частиц, агрегируется посредством домена, образующего частицы, с образованием продукта в виде частиц.
Если расстояние между С-концами слитых белков, образующих мультимер согласно изобретению, изменяется из-за связывания с доменом, образующим частицы, посредством встраивания линкера между доменом, образующим частицы, и С-концами (домена мультимеризации), то влияние на расстояние между С-концами может быть уменьшено. Линкер не имеет конкретных ограничений, но предпочтительно, он представляет собой пептид, содержащий 5-20 аминокислот, и более предпочтительно, пептид, содержащий 5-10 аминокислот. Последовательность не имеет конкретных ограничений, при условии, что она не будет ингибировать действие слитого белка на вакцину, но, например, может представлять собой линкер GS, состоящий из глицина и серина.
Линкер может быть расположен между доменом мультимеризации и доменом образования частиц мутантного белка F RSV или слитого белка, и последовательности-метки могут быть включены до и после линкера. Последовательности-метки не имеет конкретных ограничений. Однако, предпочтительно использовать последовательности меток, которые обычно применяются для очистки белка. В качестве примеров могут служить метка FLAG (SEQ ID NO: 28), His-метка (SEQ ID NO:25), Strep-метка II (SEQ ID NO:26) и т.п.
[0073]
Слитый белок, используемый для получения продукта в виде частиц, может быть получен путем связывания, с сохранением рамки считывания, ДНК, кодирующей домен, образующий частицу, а также, при необходимости, ДНК, кодирующей линкер или метку, с ДНК, кодирующей мутантный белок F RSV или слитый белок, и его экспрессии в организме хозяина. Метод экспрессии и очистки может быть осуществлен таким же методом, как и метод экспрессии и очистки мутантного белка F RSV или слитого белка.
[0074]
Конкретными примерами слитого белка, используемого для получения продукта в виде частиц, являются вариант, в котором пептид, связывающийся с каркасной частицей, связан с мутантным белком F RSV (последовательности его белков-предшественников: SEQ ID NO: 31-39, и кодирующие его полинуклеотидные последовательности: SEQ ID NO: 57-65); вариант, в котором гидрофобная пептидная цепь связана с мутантным белком F RSV (последовательности его белков-предшественников: SEQ ID NO: 44-49, и кодирующие ее полинуклеотидные последовательности: SEQ ID NO: 66-71), и вариант, в котором домен самоассоциирующегося белка связан с мутантным белком F RSV (последовательности его белков-предшественников: SEQ ID NO: 55-56, или кодирующие его полинуклеотидные последовательности: SEQ ID NO: 72-73).
Слитый белок, используемый для получения продукта в виде частиц, может содержать последовательности меток, такие как метка FLAG (DYKDDDDK: SEQ ID NO: 28) и метка (GGSDYKDDDDK: SEQ ID NO: 29), где 3 аминокислоты (GGS) присоединены к метке FLAG.
[0075]
Способ получения продукта в виде частиц
Продукт в виде частиц может быть получен путем формирования частиц с использованием полученного выше слитого белка для получения продукта в виде частиц с последующей очисткой этого продукта, если это необходимо.
При использовании каркасных частиц, стадия образования частиц может быть осуществлена путем смешивания слитых белков для получения продуктов в виде частиц с каркасными частицами, а затем удаления мутантных белков F RSV или его продуктов, которые не образуют частицы, и очистки продуктов в виде частиц. В качестве каркасных частиц могут быть использованы, например, HBsAg VLP (№ по каталогу Beacle: BCL-DC-002).
При использовании слитых белков для получения продуктов в виде частиц, содержащих гидрофобные пептидные цепи, стадия образования частиц может быть осуществлена путем связывания слитых белков для получения продуктов в виде частиц посредством гидрофобных пептидных цепей с образованием продуктов в виде частиц, а затем удаления мутантных белков F RSV или их продуктов, не образующих частиц, и очистки продуктов в виде частиц.
При использовании слитых белков для получения продуктов в виде частиц, содержащих домены самоассоциирующегося белка, стадия образования частиц может быть осуществлена путем связывания слитых белков для получения продуктов в виде частиц посредством доменов самоассоциирующегося белка с образованием продуктов в виде частиц, а затем удаления мутантных белков F RSV или их продуктов, не образующих частиц, и очистки продуктов в виде частиц.
[0076]
В продуктах в виде частиц согласно изобретению желательно, чтобы на один продукт в виде частицприходилось, например, 2 или более, предпочтительно 10 или более, а более предпочтительно 20 или более связанных тримеров.
Кроме того, в продуктах в виде частиц согласно изобретению, желательно, чтобы на 1 нм2 площади поверхности продукта в виде частиц приходилось, например, 0,0001 или более, предпочтительно 0,0005 или более, а более предпочтительно 0,001 или более связанных тримеров. В данном случае, площадь поверхности продукта в виде частиц означает площадь поверхности, если предполагается, что продукт в виде частиц представляет собой сферу, диаметр которой представляет собой теоретический диаметр, измеренный методом динамического рассеяния света. Количество частиц на площадь поверхности продукта в виде частиц может быть вычислено, например, с применением метода, в котором количество частиц на единицу продукта в виде частиц делят на площадь поверхности продукта в виде частиц.
При получении продуктов в виде частиц и выбора продуктов, которые обладают низкой способностью связывания с эпитопом I и высокой способностью связывания с эпитопом Φ, специалист в данной области может выбрать оптимальное количество частиц на один продукт в виде частиц или оптимальное количество частиц на 1 нм2 площади поверхности продукта в виде частиц.
[0077]
В настоящем изобретении, белок F RSV или мультимер, используемые для образования частиц, не ограничивается мутантным белком F RSV или мультимером согласно изобретению и не имеют конкретных ограничений при условии, что они могут быть использованы в качестве вакцины против RSV, а также может быть использован мутантный белок F RSV, отличающийся от мутантного белка F RSV согласно изобретению, или его мультимера. Так, например, могут быть использованы белки, описанные в Международной публикации № 2017/172890 или в Международной публикации № 2017/109629. То есть, технология образования частиц согласно изобретению может быть в целом использована для образования частиц белков F RSV или их мультимеров. Продукты в виде частиц согласно изобретению представляют собой продукты в виде частиц белков F RSV или их мультимеров и включают продукты в виде частиц, образованные путем агрегации двух или более белков F RSV или их мультимеров посредством доменов, образующих частицы. В качестве доменов, образующих частицы, и в качестве способа образования частиц могут быть использованы домены, образующие частицы, и способы образования частиц, описанные выше для образования частиц мутантных белков F RSV или их мультимеров.
[0078]
Фармацевтическая композиция
Другой вариант настоящего изобретения представляет собой фармацевтическую композицию, включающую экспрессионный кластер или иммуноген в качестве активного ингредиента.
Экспрессионный кластер или иммуноген может представлять собой фармацевтическую композицию для профилактики или лечения вирусной инфекции RS.
Другой вариант настоящего изобретения представляет собой вакцину RSV, которая включает экспрессионный кластер или иммуноген. Этот вариант также представляет собой вариант, в котором фармацевтическая композиция согласно вышеприведенному варианту представляет собой вакцину против RSV.
[0079]
Фармацевтическая композиция (включая вакцину против RSV, также упоминаемую далее) не имеет конкретных ограничений с точки зрения способа введения и может быть введена млекопитающим либо путем парентерального введения (например, интрадермального, внутримышечного, подкожного, трансдермального введения и введения через слизистую), либо путем перорального введения. Примерами млекопитающих являются человек, мыши, крысы, кролики, собаки, кошки, коровы, свиньи, обезьяны, шимпанзе и т.п., а предпочтительно, человек.
[0080]
Лекарственная форма фармацевтической композиции и способ ее получения хорошо известны специалистам в данной области, и фармацевтическая композиция может быть получена путем смешивания экспрессионного кластера или иммуногена с фармацевтически приемлемым носителем или т.п.
[0081]
Примерами фармацевтически приемлемого носителя являются наполнитель, лубрикант, связующее вещество и дезинтегрирующий агент в твердой композиции или растворитель, солюбилизирующий агент, суспендирующий агент, агент, придающий изотоничность, забуферивающий агент, мягчитель и т.п. в жидкой композиции. Кроме того, при необходимости, могут быть соответствующим образом использованы добавки, такие как обычный антисептик, антиоксидант, краситель, подсластитель, адсорбент и смачивающий агент в соответствующих количествах.
[0082]
Фармацевтическая композиция может включать адъювант. Примерами адъюванта являются гель, токсины бактериальных клеток, масляная эмульсия, полимерные наночастицы, синтетический адъювант, цитокин и т.п.
Примерами гелей являются гидроксид алюминия, фосфат алюминия, фосфат кальция и т.п.
Примерами токсинов бактериальых клеток являются CpG, холерный токсин, термофильный токсин Escherichia coli, коклюшный токсин, мурамилдипептид (MDP) и т.п.
Примерами масляных эмульсий являются неполный адъювант Фрейнда, MF59, SAF и т.п.
Примерами полимерных наночастиц являются иммуностимулирующие комплексы, липосомы, биоразлагаемые микросферы, QS-21, происходящий от сапонина и т.п.
Примерами синтетических адъювантов являются неионные блок-сополимеры, аналоги мурамил-пептидов, полифосфазен, синтетические полинуклеотиды и т.п.
Примерами цитокинов являются IFN-γ, IL-2, IL-12 и т.п.
[0083]
Примерами лекарственных форм для парентерального введения являются препараты для инъекций (такие как капельная инъекция, внутривенная инъекция, внутримышечная инъекция, подкожная инъекция, интрадермальная инъекция, препарат для интрацеребрального введения и препарат для интраспинального введения), препараты для наружного применения (такие как мазь, припарки и лосьоны), ингаляционные суппозитории, офтальмологические средства, глазные мази, капли в нос, ушные капли, липосомные средства и т.п.
Примерами лекарственных форм для перорального введения являются твердые или жидкие лекарственные формы, а в частности, таблетки, таблетки с покрытием, пилюли, мелкие гранулы, гранулы, порошки, капсулы, сиропы, эмульсии, суспензии, пастилки и т.п.
Фармацевтическая композиция согласно изобретению предпочтительно представляет собой жидкую лекарственную форму, а более предпочтительно, инъекцию.
[0084]
Эффективная доза фармацевтической композиции может быть определена, например, врачом исходя из различных факторов, таких как способ введения, тип заболевания, тяжесть симптомов, возраст, пол и масса тела пациента, тяжесть заболевания, фармакологические данные, такие как фармакокинетика и токсикологические признаки, независимо от того, используется ли система доставки лекарственного средства или нет, и вводится ли фармацевтическая композиция как часть комбинации с другими лекарственными средствами, или нет.
Примеры
[0085]
Ниже приводится описание Примеров. Однако, ни один из этих примеров не следует интерпретировать как ограничение объема изобретения.
[0086]
Пример 1
Образование ковалентной связи (дисульфидной связи) с использованием цистеинов способствует стабилизации структуры белка. В предыдущих исследованиях сообщалось о различных дисульфидных мутациях, которые стабилизируют пре-F-структуру в белке F RSV (далее он может упоминаться как «белок F») (Международная публикация № 2017/172890 и Международная публикация № 2017/109629). Однако, поскольку аминокислота, входящая во вторичную структуру, заменяется цистеином или образует дисульфид в отдаленной части, то существует высокий риск того, что полученный мутант может иметь структуру, отличающуюся от природной структуры (структуры перед слиянием, которую, как считается, имеет природный белок F во время его экспрессии). Поэтому, было принято решение стабилизировать структуру путем замены на цистеины аминокислотной пары, которая не имеет вторичной структуры и имеет расстояние между атомами S менее, чем 5 Å при замене на цистеины. При вычислении расстояния была использована опубликованная структурная модель (регистрационный номер PDB: 4MMS), и две произвольные аминокислоты были заменены цистеинами для получения конформации, имеющей энергетически стабильный двугранный угол N-Ca-Cb-S, и было вычислено кратчайшее расстояние между атомами S (Таблица 1). В результате, сайты мутаций мутантного белка F RSV (который в настоящем описании может называться «мутантным белком»), содержащиеся в антигенах FH_08 и FH_09, которые будут описаны позже, были перечислены в качестве кандидатов. Два эти кандидата не имеют полярной функциональной группы в боковой цепи аминокислоты перед заменой в сайте мутации, и, таким образом, они считаются превосходными также в том отношении, что отсутствует риск структурной нестабильности из-за потери взаимодействия полярной группы (водородной связи или т.п.), присутствующей в природном белке F. «L141C» в Таблице 1 означает, что лейцин в положении 141 аминокислотной последовательности SEQ ID NO: 1 был заменен цистеином. Здесь и далее в Таблице используются те же самые обозначения. Белок-предшественник (SEQ ID NO: 4) нативного антигена F представляет собой полипептид, где в указанном полипептиде (номера аминокислот 1-513 SEQ ID NO: 1), полученном путем удаления трансмембранной области и внутриклеточной области полипептида F1 из природного белка F0 штамма A2, фолдоновый домен (SEQ ID NO: 21) и последовательность (SEQ ID NO: 22), включающую последовательность распознавания тромбином, His-метку и Strep-метку II, последовательно присоединяют к С-концевой стороне полипептида, имеющего мутации P102A, I379V и M447V. В данном примере, все мутанты были созданы на основе белка-предшественника нативного антигена F и, таким образом, имеют мутации P102A, I379V и M447V. Белок-предшественник нативного F подвергают процессингу во время экспрессии, что приводит к удалению сигнального пептида и области pep27.
[0087]
Таблица 1
[0088]
Нативный антиген F получают путем экспрессии полинуклеотида (SEQ ID NO: 5), кодирующего белок-предшественник (SEQ ID NO: 4) нативного антигена F, с использованием клетки млекопитающего в качестве хозяина. Нативный антиген F образует четвертичную структуру после того, как белок-предшественник (SEQ ID NO: 4) нативного антигена F подвергается процессингу во время экспрессии. В результате процессинга, сигнальный пептид и область pep27 удаляются, а оставшаяся последовательность полипептида F2 и последовательность, включающая полипептид F1 (внеклеточная область), фолдоновый домен, последовательность распознавания тромбином, His-метку и Strep-метку II связаны дисульфидной связью, и, кроме того, ожидается, что гомотример будет образовываться за счет силы связывания, в основном создаваемой фолдоновым доменом.
Конкретный способ получения нативного антигена F показан ниже. Указанный выше полинуклеотид (SEQ ID NO: 5) встраивали в вектор pcDNA3.4 (Thermo Fisher Scientific, Inc.) с применением стандартной методики генной инженерии. Встраивание последовательности ДНК-мишени было подтверждено в анализе последовательности оснований, и был сконструирован экспрессионный вектор клеток млекопитающих, экспрессирующий белок-предшественник нативного антигена F. Вектор для введения в клетку очищали стандартным методом очистки плазмидного вектора.
[0089]
Экспрессию нативного антигена F осуществляли посредством экспрессии секретируемого белка в клетках млекопитающих с использованием экспрессионной системы Expi293 (Thermo Fisher Scientific, Inc.). Очищенный вектор вводили в клетку Expi293 с использованием реагента Expi Fectamin 293, который представляет собой реагент для переноса генов из того же набора, а затем кодируемый нативный антиген F был секретирован и экспрессирован путем культивирования в течение приблизительно 5 дней, и только компонент супернатанта был получен путем центрифугирования и использованием фильтра с диаметром отверстий 0,22 мкм.
[0090]
Очистку нативного антигена F проводили с помощью аффинной хроматографии на высокоэффективной колонке с Ni-сефарозой (GE Healthcare, Inc.). Ni-носитель подвергали взаимодействию с вышеуказанным компонентом культурального супернатанта, разведенным приблизительно 3 раза буфером для связывания (20 ммоль/л трис-HCl, pH 7,4, 500 ммоль/л NaCl, 20 ммоль/л имидазола) в течение суток, а затем его наносили на элюирующий буфер (20 ммоль/л Tris-HCl, pH 7,4, 500 ммоль/л NaCl, 500 ммоль/л имидазола) для элюирования каждого антигена. После этого, с помощью ультрафильтрационной мембраны проводили замену растворителя на PBS (pH 7,4), и продукт использовали в качестве нативного антигена F.
[0091]
Полинуклеотид, кодирующий белок-предшественник антигена FH_08, где двойная мутация L141C и L373C была введена в белок-предшественник нативного антигена F, получали так же, как описано выше. Далее таким же образом, как описано выше, был получен полинуклеотид, кодирующий белок-предшественник антигена FH_09, где двойная мутация L142C и L373C была введена в белок-предшественник нативного антигена F. Эти белки экспрессировали и очищали с применением описанного выше метода получения антигенов FH_08 и FH_09.
[0092]
Связывающую способность каждого антигена с каждым антителом оценивали с использованием Octet QK384 (ForteBio, Inc.) в условиях PBS (pH 7,4), 25°C и 1000 об/мин. Каждое антитело связывали с сенсорным чипом путем нанесения каждого раствора антитела на 180 секунд на сенсорный чип с белком А (ForteBio, Inc.), уравновешенный PBS (pH 7,4), и увеличение детектируемого значения, если каждый раствор антигена последовательно наносили в течение 180 секунд, определяли как значение сигнала связывания каждого антигена с каждым антителом. В качестве антитела, нанесенного на сенсорный чип, использовали антитело D25, способное специфически обнаруживать эпитоп Φ. Величину сигнала связывания нормализовали путем определения отношения к величине сигнала связывания при использовании паливизумаба (внутримышечная инъекция Synagis, AbbVie, Inc.), способного к связыванию независимо от конформационного изменения F-антигена.
[0093]
С помощью описанной выше процедуры был получен результат, свидетельствующий о том, что антигены FH_08 и FH_09, содержащие мутантные белки, в которых цистеиновые точковые мутации были введены в двух сайтах для образования дисульфидной связи, значительно улучшали стабильность эпитопа Φ при хранении по сравнению с нативным антигеном F, содержащим белок F до введения мутаций (Фиг. 1).
[0094]
Пример 2
Имеется сообщение о том, что природный белок F сначала расщепляется в первом сайте распознавания фурином с образованием мономерной структуры, а затем расщепляется во втором сайте распознавания фурином с удалением области pep27, что тем самым способствует тримеризации (Anders Krarup et al., 2015, Nature communications 6:8143). Однако, поскольку область pep27 в высокой степени консервативна по длине последовательности и по типу аминокислот среди штаммов вируса, выделенных из природного источника, то считается, что область pep27 может оказывать незаменимое влияние на формирование стерической структуры антигена. Поэтому, во второй сайт распознавания фурином была введена мутация для ингибирования расщепления области pep27, и было исследовано влияние мутации на образование тримера. Содержание мутаций белков-предшественников антигенов, полученных как описано ниже, было систематизировано в Таблице 2. «FH_08+R135N, R136N» в Таблице 2 означает, что в белке-предшественнике антигена FH_08, аргинин в положении 135 в аминокислотной последовательности SEQ ID NO: 1 был заменен аспарагином, и аргинин в положении 136 был заменен аспарагином. «FH_09+R135N, R136N» означает, что в белке-предшественнике антигена FH_09, аргинин в положении 135 в аминокислотной последовательности SEQ ID NO: 1 был заменен аспарагином, и аргинин в положении 136 был заменен аспарагином. В антигенах FH23-25, последовательность pep27 (p27) в белке-предшественнике антигена FH08 была заменена различными линкерами GS. В качестве GS-линкеров использовали GSGSGS (SEQ ID NO: 18), (GGGGS)2 (SEQ ID NO: 19), (GGGGS)3 (SEQ ID NO: 20).
[0095]
Таблица 2
[0096]
Полинуклеотид, кодирующий каждый из белков-предшественников антигенов, встраивали в вектор pcDNA3.4 (Thermo Fisher Scientific, Inc.) с применением стандартной методики генной инженерии. Встраивание последовательности ДНК-мишени было подтверждено с помощью анализа последовательности оснований, и конструировали каждый из экспрессионных векторов клеток млекопитающих, экспрессирующих белки-предшественники антигенов. Вектор для введения в клетку очищали с применением стандартного метода очистки плазмидного вектора.
[0097]
Экспрессию и очистку каждого антигена проводили с помощью процедуры, описанной в Примере 1.
Способность к связыванию каждого антигена с каждым антителом также оценивали с помощью процедуры, описанной в Примере 1. Однако, в качестве антитела для нанесения на сенсорный чип добавляли антитело AM14, способное обнаруживать образование тримера pre-F. Величину сигнала связывания антитела D25 и антитела AM14 нормализовали путем определения соотношения относительно значения сигнала связывания в случае использования антитела Synagis (AbbVie, Inc., внутримышечная инъекция Synagis), способного связываться независимо от конформационного изменения F-антигена.
[0098]
С помощью описанной выше процедуры был получен результат, указывающий на то, что антигены FH_21, FH_24 и FH_25, содержащие мутантные белки, в каждом из которых мутация была добавлена во второй сайт распознавания фурином, улучшали образование тримеров с сохранением стабильности эпитопа Φ при хранении по сравнению с антигеном FH_08 до введения мутации. Среди них, улучшение образования тримера было наиболее заметно в антигене FH_21, который расположен ближе к области pep27 нативного антигена F по сравнению с антигенами (FH_24 и FH_25), в которых область pep27 была заменена искусственным линкером (Фиг. 2 и 3).
[0099]
Пример 3
Аминокислоты в положениях 189 и 190 белка-предшественника нативного антигена F представляют собой треонин и серин и являются гидрофильными аминокислотами, поскольку каждая из них имеет гидроксильную группу в боковой цепи. Однако, аминокислоты, расположенные стерически более близко к аминокислотам в положениях 189-190, представляют собой изолейцин в положении 57, изолейцин в положении 167, лейцин в положении 171, валин в положении 179 и лейцин в положении 260 и т.п. Однако, поскольку все они являются гидрофобными аминокислотами, то считается, что структурная стабильность может быть нарушена. Поэтому рассматривается возможность структурной стабилизации и стимуляции повышения уровня экспрессии путем замены аминокислот в положениях 189-190 на гидрофобные аминокислоты (валин, лейцин, изолейцин). Описание мутаций в белках-предшественниках антигенов, полученных как описано ниже, систематизировано в Таблице 3. «FH_21+T189I» в Таблице 3 означает, что в белке-предшественнике антигена FH_21, треонин в положении 189 в аминокислотной последовательности SEQ ID NO: 1 заменен на изолейцин. «FH_21+T189V» означает, что в белке-предшественнике антигена FH_21, треонин в положении 189 в аминокислотной последовательности SEQ ID NO: 1 заменен на валин. «FH_21+T189I, S190V» означает, что в белке-предшественнике антигена FH_21, треонин в положении 189 в аминокислотной последовательности SEQ ID NO: 1 заменен на изолейцин, а серин в положении 190 заменен на валин. «FH_21+T189L, S190V» означает, что в белке-предшественнике антигена FH_21, треонин в положении 189 в аминокислотной последовательности SEQ ID NO: 1 заменен на лейцин, а серин в положении 190 заменен на валин. «FH_21+T189V, S190V» означает, что в белке-предшественнике антигена FH_21, треонин в положении 189 в аминокислотной последовательности SEQ ID NO: 1 заменен на валин, и серин в положении 190 заменен на валин. «FH_50+K42R, V384T» означает, что в белке-предшественнике антигена FH_50, лизин в положении 42 в аминокислотной последовательности SEQ ID NO: 1 заменен на аргинин, а валин в положении 384 заменен на треонин. Кроме того, были удалены точковые мутации, встречающиеся в природных изолятах вируса, и была исследована каждая отдельная мутация. Был подтвержден эффект повышения уровня экспрессии при двух точковых мутациях (K42R, V384T), и, таким образом, исследование проводили путем введения такой мутации.
[0100]
Таблица 3
[0101]
Полинуклеотид, кодирующий каждый из белков-предшественников антигенов, встраивали в вектор pcDNA3.4 (Thermo Fisher Scientific, Inc.) с применением стандартной методики генной инженерии. Встраивание последовательности ДНК-мишени подтверждали с помощью анализа последовательности оснований, и конструировали каждый из экспрессионных векторов клеток млекопитающих, экспрессирующих белки-предшественники антигенов. Вектор для введения в клетку очищали с применением стандартного метода очистки плазмидного вектора.
[0102]
Экспрессию и очистку каждого из антигенов проводили с помощью процедуры, описанной в Примере 2.
Уровень экспрессии каждого антигена оценивали путем измерения уровня поглощения на стандартном спектрофотометре. Уровень экспрессии y (y=A280xM/εxVp/Vc) на объем экспрессионной культуры вычисляли по поглощению (на длине волны 280 нм) A280 очищенного раствора антигена, по молярному коэффициенту поглощения ε и по молекулярной массе M, которые оценивали исходя из информации о первичной аминокислотной последовательности, по объему раствора антигена Vp после очистки и по объему экспрессионной культуры Vc.
[0103]
Способность связывания каждого антигена с антителом D25 и антителом AM14 оценивали с помощью процедуры, описанной в Примере 2.
С помощью вышеописанной процедуры был получен результат, показывающий, что антигены FH_50, FH_51, FH_52, FH_53 и FH_55, в каждый из которых была внесена мутация, а именно, замена гидрофобными аминокислотами (V, I, L) в положениях 189-190, значительно повышали уровень экспрессии с сохранением стабильности эпитопа Ф при хранении и образовании тримера по сравнению с антигеном FH_21 до введения мутации. Кроме того, был получен результат, показывающий, что антиген FH_82, в который были добавлены две точковые мутации (K42R, V384T), полученные из естественных мутаций, которые могут встречаться в природных вирусах, также значительно повышал уровень экспрессии с сохранением стабильности эпитопа Ф при хранении и образовании тримера (Фиг. 4-6).
[0104]
Пример 4
Аминокислота в положении 60 белка-предшественника нативного F представляет собой глутаминовую кислоту и является кислотной аминокислотой, и хорошо известно, что она является отрицательно заряженной в условиях, когда растворитель является нейтральным. Эта аминокислота стерически примыкает к аспарагиновой кислоте в положении 194, которая также является отрицательно заряженной в нейтральных условиях, а поэтому существует возможность структурной нестабильности из-за близости отрицательных зарядов. Следовательно, в аминокислотную последовательность белка-предшественника нативного F была введена мутация, а именно, замена аминокислоты в положении 60 на аминокислоту, не являющуюся кислотной аминокислотой. Однако, при замене аминокислоты в положении 60 на аспарагин для предупреждения превращения аминокислоты в положениях 60-62 в хорошо известный мотив связывания сахарной цепи N-типа (Asn-X-Ser/Thr), была одновременно проведена замена серина в положении 62 на аланин. Описание мутаций в белках-предшественниках антигенов, полученных как описано ниже, систематизировано в Таблице 4.
[0105]
Таблица 4
[0106]
Полинуклеотид, кодирующий каждый из белков-предшественников антигенов, встраивали в вектор pcDNA3.4 (Thermo Fisher Scientific, Inc.) с применением стандартной методики генной инженерии. Встраивание последовательности ДНК-мишени подтверждали с помощью анализа последовательности оснований, и конструировали каждый из экспрессионных векторов клеток млекопитающих, экспрессирующих белки-предшественники антигенов. Вектор для введения в клетку очищали с применением стандартного метода очистки плазмидного вектора.
[0107]
Экспрессию и очистку каждого из антигенов проводили с помощью процедуры, описанной в Примере 2.
Способность связывания каждого антигена с антителом D25 и антителом AM14 оценивали с помощью процедуры, описанной в Примере 2.
С помощью вышеописанной процедуры был получен результат, показывающий, что стабильность эпитопа Φ при хранении и образование тримера улучшались, если в положение 60 были введены замены на M, F, L, S и T (фиг. 7 и 8).
[0108]
Пример 5. Сравнение с предшественником
Одной из наиболее широко известных мутаций, вводимых в качестве метода стабилизации пре-белка типа F антигена F, является мутация DS-Cav1 (S155C, S290C, S190F, V207L) (Jason S. McLellan et al., 2013, Science 342:592). Таким образом, антигены, представленные в Таблице 5, в которых полезные мутации были дополнительно введены в нужный антиген FH_50 и антиген FH_50, описанные в вышеприведенных примерах, сравнивали с антигеном F_DS-Cav1, в который была введена мутация DS-Cav1. Кроме того, был также получен антиген ΔFP furinwt Fecto (Kurt A. Swanson et al., 2014, Journal of Virology, 88 (20): 11802), который, как известно, имеет пост-F-тримерную структуру, и этот антиген был использован для оценки антигенов. Описание мутаций в белках-предшественниках антигенов, полученных как описано ниже, систематизировано в Таблице 5. Каждый из белков-предшественников антигенов FH_81-85 был получен путем введения мутации E60M, K42R+V384T или E60M+K42R+V384T в белок-предшественник антигена FH_50.
[0109]
Таблица 5
[0110]
Полинуклеотид, кодирующий каждый из белков-предшественников антигенов, встраивали в вектор pcDNA3.4 (Thermo Fisher Scientific, Inc.) с применением стандартной методики генной инженерии. Белок-предшественник антигена F_DS-Cav1 представляет собой аминокислотную последовательность, состоящую из SEQ ID NO: 23, а белок-предшественник ΔFP furinwt Fecto представляет собой аминокислотную последовательность, состоящую из SEQ ID NO: 24. Встраивание последовательности ДНК-мишени было подтверждено с помощью анализа последовательности оснований, и был сконструирован каждый из экспрессионных векторов клеток млекопитающих, экспрессирующих белки-предшественники антигенов. Вектор для введения в клетку очищали с применением стандартного метода очистки плазмидного вектора.
[0111]
Экспрессию и очистку каждого из антигенов проводили с помощью процедуры, описанной в Примере 2.
Уровень экспрессии каждого антигена оценивали с помощью процедуры, описанной в Примере 3.
[0112]
Тест на способность связываться с антителом
Способность к связыванию каждого антигена с каждым антителом оценивали с помощью процедуры, описанной в Примере 2. Однако, в качестве антитела для нанесения на сенсорный чип добавляли антитело 131-2А, способное распознавать эпитоп I. Величину сигнала связывания антитела D25 и антитела AM14 и антитела 131-2А нормализовали путем определения его отношения к значению сигнала связывания в случае использования антитела Synagis (AbbVie, Inc., внутримышечная инъекция Synagis), способного связываться независимо от конформационного изменения антигена F.
[0113]
С помощью вышеописанной процедуры был получен результат, показывающий, что для антигенов FH_50, FH_81, FH_82 и FH_85 по сравнению с антигеном F_DS-Cav1, описанным в литературе, несмотря на отсутствие значительного изменения стабильности при хранении эпитопа Φ и образовании тримера, эпитоп I, который, как известно, индуцирует антитела, обладающие довольно низкой способностью нейтрализовать вирусную инфекцию, практически не обнаруживался (фиг. 9-11). Это позволяет предположить, что хотя вакцина ассоциируется с риском обострения симптомов в результате активации иммунной клетки, которая в небольшой степени способствует нейтрализации вируса, однако, такой риск снижается при введении вновь обнаруженных мутаций по сравнению с риском при введении мутации DS-Cav1.
[0114]
Тест на адсорбцию антител, нейтрализующих вирус, в сыворотке человека
В результате проведения вышеописанной процедуры было показано, что существует вероятность того, что антигены FH_50, FH_81, FH_82, FH_85 обладают эффектом подавления индуцирования иммунитета, который не способствует нейтрализации вируса. Для более подробного изучения такого эффекта с использованием сыворотки здоровых взрослых людей, было проверено, с какими типами антител в сыворотках здоровых взрослых связываются антигены FH_50, FH_81, FH_82, FH_85 и F_DS-Cav1.
[0115]
Сыворотки трех здоровых взрослых индивидуумов подвергали процедуре адсорбции антигеном с использованием магнитных шариков Dynabeads с последующим выделением His-метки и получением нужного продукта (Thermo Fisher Scientific, 10103D). Сначала наносили 20 мкг антигена F_DS-Cav1, FH_50, FH_81, FH_82 или FH_85 и стимулировали связывание с шариками в 45 мкл раствора, дважды промытыми PBS (рН 7,4), а затем блокировали альбумином бычьей сыворотки. Затем 100 мкл сыворотки каждого здорового взрослого человека 10-кратно разводили PBS (pH 7,4), а затем добавляли шарики и смесь инкубировали (4°C, 1 час, при встряхивании), после чего антитела, связанные с антигенами, удаляли путем удаления шариков.
[0116]
Был проведен ELISA-анализ, в котором был иммобилизован антиген ΔFP furinwt Fecto, для подтверждения уровня связывания антитела (антитела, которое, как предполагается, обладает относительно низкой способностью нейтрализовать вирусную инфекцию) с пост-F. В качестве антигенсвязывающего буфера использовали карбонатно-бикарбонатный буфер (Sigma-Aldrich, C3041-50CAP), а для разведения образца и промывки планшета использовали TBS (TaKaRa, T9142). 2,0 пмоль антигена ΔFP furinwt Fecto добавляли в половину лунок 96-луночного планшета для прямой иммобилизации и блокирования альбумином бычьей сыворотки. Серию 4-кратных разведений приготавливали путем соответствующего добавления буфера к сыворотке здорового взрослого человека или к сыворотке здорового взрослого человека после проведения процедуры адсорбции антигеном, а затем добавляли в планшет и инкубировали (при комнатной температуре, 1 час, в стационарных условиях). После этого, разведенную сыворотку удаляли и подвергали связыванию с конъюгированным с пероксидазой антителом против человеческого IgG (Jackson Immuno Research, 709-035-149), а затем проявляли окраску с помощью стандартного реагента для обнаружения пероксидазы, после чего, интенсивность проявления окраски определяли путем измерения поглощения на стандартном планшет-ридере. Зависимость между скоростью разведения сыворотки и поглощением вычисляли путем кусочно-линейной интерполяции между точками измерения, и обратную величину степени разведения, при котором поглощение равно 0,2, определяли как титр антитела, связывающегося с пост-F, и относительную величину снижения титра сывороточных антител после процедуры адсорбции, если титр сывороточных антител до процедуры адсорбции составлял 100%, определяли как скорость адсорбции (фиг. 12).
[0117]
Для того, чтобы подтвердить, в какой степени антитело, обладающее способностью нейтрализовать вирусную инфекцию, было удалено при проведении процедуры адсорбции, оценку инфекции проводили с использованием вируса RS (ATCC, VR-1540) и клеток Vero (ATCC, CCL-81). Серию 5-кратных разведений приготавливали путем соответствующего добавления PBS (pH 7,4) к сыворотке здорового взрослого человека или к сыворотке здорового взрослого человека после процедуры адсорбции антигеном, а затем смешивали с вирусом RS и инкубировали (при комнатной температуре, 30 минут, в стационарных условиях). после чего в каждую лунку 96-луночного планшета высевали 4×104 клеток, добавляли к клеткам Vero, культивировали в течение 1 дня, и инкубировали (37°C, в условиях влажности, 2 часа, в стационарных условиях). После этого, раствор вируса удаляли, а затем культуральную среду, включая DMEM и высокую концентрацию глюкозы (Thermo Fisher Scientific, 11965), в которую раствор пирувата натрия (Nakarai, 06977-34) и смешанный маточный раствор антибиотиков и противогрибковых препаратов (Nakarai, 09366-44) были добавлены в количестве 1% (об./об.), добавляли в концентрации 100 мкл на лунку и дополнительно инкубировали (37°C, в условиях влажности, 16 часов, в стационарных условиях). После этого, клетки иммобилизовали параформальдегидом, и для клеток, пермеабилизированных Тритоном Х-100 (Nakarai, 35501-15), ядра инфицированных клеток и всех клетки окрашивали с использованием антител против RSV, слитых белков, всех штаммов типа А, В, клона 133-1H, Alexa Fluor 488 (Merck, MAB8262X) и Heochst33258 (Dojindo, 343-07961), и количество ядер инфицированных клеток подсчитывали на визуализирующем сканере IN для клеток (GE Healthcare) в целях определения числа инфицированных вирусом клеток. Взаимосвязь между степенью разведения Х каждой сыворотки и числом инфекций Y определяли путем построения сигмовидной кривой (y=Ymax/(1+(x/IC50)C)) по экспериментальным результатам (полученным способом вычисления погрешностей с применением методов наименьших квадратов), а в качестве титра, нейтрализующего вирусную инфекцию, использовали обратную величину IC50. Если титр, нейтрализующий вирусную инфекцию, в сыворотке перед процедурой адсорбции был принят за 100%, то относительную величину снижения титра, нейтрализующего вирусную инфекцию, после проведения процедуры адсорбции, принимали за скорость адсорбции (Фиг. 13).
[0118]
С помощью вышеописанной процедуры было показано, что по сравнению с антигеном F_DS-Cav1, описанным в литературе, антигены FH_50, FH_81, FH_82 и FH_85 обладают низкой способностью связываться с антителом, распознающим пост-F, присутствующим в сыворотке здорового взрослого человека, и только приблизительно половина антител могли быть связаны в процессе адсорбции. Тем не менее, было также показано, что почти все антитела, которые вносят вклад в способность нейтрализовать вирусную инфекцию, связываются так же, как и F_DS-Cav1, а антитела, оставшиеся после процедуры адсорбции, почти не обладают способностью нейтрализовать вирусную инфекцию. Приведенные выше результаты позволяют предположить, что по сравнению с введением мутации DS-Cav1, вновь обнаруженная мутация обладает меньшей способностью связываться с антителом, которое является неэффективным для нейтрализации вирусной инфекции, и при использовании этой мутации в качестве вакцинного антигена будет снижаться риск индуцирования антитела, которое не способствует защите от вирусной инфекции.
[0119]
Подтверждение антител, индуцированных мышиным иммунитетом
Было высказано предположение, что с помощью описанной выше процедуры, мутантный белок будет обладать эффектом подавления вырабатывания иммунитета, который не способствует нейтрализации вируса. Для более детального изучения этого эффекта был проведен анализ антител, индуцированных посредством иммунизации мышей антигеном FH_50, FH_81, FH_82, FH_85 или F_DS-Cav1.
[0120]
Иммунные сыворотки мышей получали путем иммунизации BALB/cAnNCrlCrlj (Japanese Charles River) (самок в возрасте 7 недель) мутантными белками. Раствор, полученный путем тщательного смешивания антигена FH_50, FH_81, FH_82, FH_85 или F_DS-Cav1 (концентрация антигена: 0,80 мг/мл), растворенного в PBS (pH 7,4), и гидроксида алюминия (InvivoGen, vac-alu-250), в равных количествах вводили внутримышечно в количестве 50 мкл в задние отделы бедра мышей на 0-й день иммунизации и на 21-й день иммунизации (по 5 мышей в группе). Кровь получали из задней полой вены на 42-й день иммунизации, и полученную кровь инкубировали (при комнатной температуре, приблизительно 2 часа, в стационарных условиях), а затем сыворотку собирали путем центрифугирования и инактивировали (55°С, 30 минут) для приготовления иммунной сыворотки мышей.
[0121]
Чтобы подтвердить соотношение, при котором индуцируются антитела, которые связываются с конформациями после слияния, каждую мышиную иммунную сыворотку подвергали адсорбции антигеном с использованием магнитных шариков Dynabeads с последующим выделением His-метки и получением нужного продукта (Thermo Fisher Scientific, 10103D). Сначала наносили 20 мкг антигена ΔFP furinwt Fecto, и стимулировали связывание с шариками в 45 мкл раствора, дважды промытыми PBS (рН 7,4), а затем блокировали альбумином бычьей сыворотки. Затем 50 мкл каждой иммунной мышиной сыворотки 20-кратно разводили PBS (pH 7,4), а затем добавляли шарики и смесь инкубировали (4°C, 1 час, при встряхивании), после чего антитела, связанные с антигенами, удаляли путем удаления шариков.
[0122]
Затем был проведен ELISA-анализ, в котором был иммобилизован антиген F_DS-Cav1, и оценивали степень снижения уровня связывания антитела против антигена F с помощью процедуры адсорбции антигеном. В качестве антигенсвязывающего буфера использовали карбонатно-бикарбонатный буфер (Sigma-Aldrich, C3041-50CAP), а для разведения образца и промывки планшета использовали TBS (TaKaRa, T9142). 2,0 пмоль антигена F_DS-Cav1 добавляли в половину лунок 96-луночного планшета для прямой иммобилизации и блокирования альбумином бычьей сыворотки. Серию 5-кратных разведений приготавливали путем соответствующего добавления буфера к мышиной иммунной сыворотке или к мышиной иммунной сыворотке после адсорбции антигеном, а затем добавляли в планшет и инкубировали (при комнатной температуре, 2 часа). После этого, разведенную сыворотку удаляли и подвергали связыванию с конъюгированным с пероксидазой антителом против мышиного антигена (DaKo, P0161), а затем проявляли окраску с помощью стандартного реагента для обнаружения пероксидазы, после чего, интенсивность проявления окраски определяли путем измерения поглощения на стандартном планшет-ридере. Зависимость между скоростью разведения сыворотки и поглощением вычисляли путем кусочно-линейной интерполяции между точками измерения, и обратную величину степени разведения, при котором поглощение равно 0,2, определяли как титр детектируемого антитела против антигена F, и относительную величину снижения титра сывороточных антител после процедуры адсорбции, если титр сывороточных антител до процедуры адсорбции составлял 100%, определяли как скорость адсорбции (Фиг. 14).
[0123]
С помощью вышеописанной процедуры было показано, что по сравнению с антигеном F_DS-Cav1, описанным в литературе, антигены FH_50, FH_81, FH_82 и FH_85 обладают низкой способностью индуцировать антитело, распознающее пост-F. То есть, был получен результат, свидетельствующий о том, что при использовании антигенов FH_50, FH_81, FH_82, FH_85 в качестве вакцинных антигенов, риск индуцирования антитела, не способствующего защите от вирусной инфекции, является низким.
[0124]
Пример 6. Второе сравнение с предшественником
Все фрагменты белка F антигенов FH_50, FH_81, FH_82 и FH_85, полученные как описано в Примере 5, содержат мутации L141C и L373C. Эти мутации представляют собой мутации, созданные для введения дисульфидной связи как описано в Примере 1. Однако, в более раннем патенте (Международной публикации № 2017/109629) оценивали введение дисульфидной связи в относительно близкий сайт (идентификатор мутанта: pXCS524.L142C, и N371C) и оптимизированные антигены с дополнительными мутациями (ID мутанта: pXCS881.L142C, N371C, S55C, L188C, T54H, V296I, D486S, E487Q и D489S). Поэтому, полезные антигены, полученные как описано выше в Примерах, сравнивали с антигеном Pf524 или Pf881, в которые была введена мутация pXCS524 или мутация pXCS881. Описание мутаций в белках-предшественниках антигенов, полученных как описано ниже, систематизировано в Таблице 6.
[0125]
Таблица 6
[0126]
В качестве антигенов FH_08, FH_82 и FH85 использовали антигены, описанные в Примере 1, в Примере 3 и в Примере 5, соответственно. Каждый из полинуклеотидов, кодирующих белки-предшественники Pf524 и Pf881, встраивали в вектор pcDNA3.4 (Thermo Fisher Scientific, Inc.) с применением стандартного метода генной инженерии, и такое встраивание последовательности ДНК-мишени было подтверждено с помощью анализа последовательности оснований, в результате чего был сконструирован каждый из экспрессионных векторов клеток млекопитающих, экспрессирующих белки-предшественники антигенов. Вектор для введения в клетку очищали с применением стандартного метода очистки плазмидного вектора.
[0127]
Экспрессию и очистку каждого из антигенов проводили с помощью процедуры, описанной в Примере 2.
Уровень экспрессии каждого антигена оценивали с помощью процедуры, описанной в Примере 3.
[0128]
Тест на способность связываться с антителом
Способность каждого антигена связываться с каждым антителом оценивали с помощью процедуры, описанной в Примере 5. Однако, значение сигнала связывания устанавливали равным значению, полученному путем вычитания детектируемого значения, если PBS (рН 7,4) наносили в течение 180 секунд, из детектируемого значения при нанесении каждого раствора антигена в течение 180 секунд (Фиг. 15-17).
[0129]
С помощью описанной выше процедуры был получен результат, показывающий, что для антигена FH_08 по сравнению с антигеном Pf524, в который была введена мутация, описанная в более раннем патенте, в то время как стабильность при хранении эпитопа Φ и образование тримера являются превосходными, эпитоп I, который, как известно, индуцирует антитело, обладающей довольно низкой способностью нейтрализовать вирусную инфекцию, практически не обнаруживается. Кроме того, был получен результат, показывающий, что аналогичным образом для оптимизированных антигенов FH_82 и FH_85 по сравнению с антигеном Pf881, в который была введена мутация, описанная в более раннем патенте, в то время как стабильность при хранении эпитопа Φ и образование тримера являются превосходными, эпитоп I, который, как известно, индуцирует антитело, обладающее довольно низкой способностью нейтрализовать вирусную инфекцию, практически не обнаруживается. Это позволяет предположить, что полезные мутации с введением дисульфида, описанные в приведенном выше примере, представляют собой мутации, которые предпочтительно индуцируют группу антител, которые, как предполагается, дают более сильный эффект в отношении нейтрализации вируса по сравнению с мутациями с введением дисульфида (L142C и N371C), описанными в более раннем патенте.
[0130]
Подтверждение антител, индуцированных мышиным иммунитетом
На основании вышеприведенного эксперимента было высказано предположение, что антигены FH_82 и FH_85 индуцируют иммунитет, который не способствует нейтрализации вируса, в меньшей степени, чем у мутанта Pf881, в который была введена мутация как описано в более раннем патенте. Для более детального изучения этого эффекта был проведен анализ антител, индуцированных путем иммунизации мышей антигеном FH_82, FH_85 или Pf881.
[0131]
Анализ введенных антител проводили в соответствии с процедурой, описанной в Примере 5, и измеряли скорость продуцирования титра антител с помощью процедуры адсорбции антигеном ΔFP furinwt Fecto (фиг. 18).
[0132]
При проведении вышеописанной процедуры было показано, что по сравнению с антигеном Pf881, в который была введена мутация как описано в более раннем патенте, антигены FH_82 и FH_85 давали более низкий уровень индуцирования антитела, распознающего пост-F. То есть, этот результат позволяет предположить, что при использовании антигенов FH_82 и FH_85 в качестве вакцинных антигенов предпочтительно индуцируется группа антител, которые, как считается, обладают более сильным эффектом в отношении нейтрализации вируса по сравнению с Pf881.
[0133]
Пример 7. Получение частиц антигена с использованием каркасных частиц
Для дальнейшего снижения риска индуцирования антител, которые не способствуют защите от вирусной инфекции, может оказаться эффективным уменьшение, за счет эффекта стерических затруднений, доступности антител или различных иммунных клеток к антигенному сайту, распознаваемому антителом, которое распознает пост-F, такое как антитело, распознающее эпитоп I. Поэтому рассматривается возможность того, что эффект стерических затруднений может быть достигнут путем иммобилизации антигенов на соответствующих каркасных частицах, так чтобы сайт распознавания пост-F-распознающего антитела находился на внутренней стороне.
Однако, если антигены презентированы на подходящих каркасных частицах, если плотность является низкой, то есть, если промежутки между антигенами равны или превышают определенную длину, или если между антигенами и каркасными частицами встроены гибкие линкеры, имеющие определенную длину или больше, то считается, что описанный выше эффект стерических затруднений не может быть достигнут. Таким образом, линкеры GS различной длины оценивали с использованием VLP HBsAg, которые, как считается, способны обеспечивать презентацию антигена с относительно высокой плотностью.
[0134]
Приготовление частиц антигена
В качестве каркасных частиц использовали бионанокапсулу (Bionanocapsule-ZZ) (Beacle Inc., BCL-DC-002), способную связываться со слитым белком Fc.
В качестве белков-предшественников антигенов использовали белок-предшественник (SEQ ID NO: 31) антигена F_DS-Cav1-Fc, белок-предшественник (SEQ ID NO: 32) FH_82-Fc и белок-предшественник (SEQ ID NO: 33) FH_85-Fc, в каждом из которых, последовательность (SEQ ID NO: 22), содержащая последовательность распознавания тромбином, His-метку, Strep-метку II в белках-предшественниках F_DS-Cav1, FH_82 и FH_85, описанных выше, была заменена последовательностью Fc (SEQ ID NO: 30). Кроме того, что касается белка-предшественника антигена F_DS-Cav1-Fc и белка-предшественника FH_85-Fc, то эти белки, в которых линкер GS (GGGGSGGGGSGGGGSGGGGS (SEQ ID NO: 75), GGGGSGGGGS (SEQ ID NO: 19) или GGGGS (SEQ ID NO: 27)) встраивали непосредственно перед последовательностью Fc, были получены на основе соответствующих белков-предшественников (SEQ ID NO: 34-39) с применением нижеследующего метода.
[0135]
Полинуклеотид, кодирующий каждый из вышеуказанных белков-предшественников, встраивали в вектор pcDNA3.4 (Thermo Fisher Scientific, Inc.) с применением стандартного метода генной инженерии. Встраивание последовательности ДНК-мишени было подтверждено с помощью анализа последовательности оснований, и был сконструирован каждый из экспрессионных векторов клеток млекопитающих, экспрессирующих белки-предшественники антигенов. Вектор для введения в клетку очищали с применением стандартного метода очистки плазмидного вектора.
[0136]
Экспрессию белка-предшественника каждого антигена осуществляли посредством экспрессии секретируемого антигена клеток млекопитающих с использованием экспрессионной системы Expi293 (Thermo Fisher Scientific, Inc.). Каждый очищенный вектор вводили в клетку Expi293 с использованием реагента Expi Fectamin 293, который представляет собой реагент для переноса генов из того же набора, и каждый кодируемый антиген секретировали и экспрессировали путем культивирования в течение приблизительно 4-5 дней, а компонент супернатанта получали путем центрифугирования и с использованием фильтра с диаметром отверстий 0,22 мкм.
Очистку каждого антигена проводили с применением стандартного метода аффинной очистки на колонке для очистки с белком А. После очистки проводили замену растворителя на PBS (рН 7,4) с использованием ультрафильтрационной мембраны.
Каждый из приготовленных антигенов смешивали с VLP HBsAg в соотношении приблизительно 135 молекул (приблизительно 45 молекул в виде тримера) на одну частицу VLP HBsAg в среднем и иммобилизовали на каркасных частицах путем выдерживания смеси при комнатной температуре в течение одного часа. В этом случае, почти полное отсутствие неиммобилизованного антигена было подтверждено путем проведениия электрофореза в нативном ПААГ с окрашиванием синим красителем.
[0137]
Антигены, используемые в анализе, описанном в этом Примере, и антигены в виде частиц, полученные путем иммобилизации антигенов на каркасных частицах (которые далее могут также называться «антигенами в виде частиц»), систематизированы в Таблице 7.
[0138]
[Таблица 7]
[0139]
Тест на способность связываться с антителом
Способность F_DS-Cav1, F_DS-Cav1-VLP, FH_82, FH_82-VLP, FH_85 и FH_85-VLP, которые представляют собой антигены или антигены в виде частиц, связываться с антителами оценивали с помощью процедуры, описанной в Примере 6 (Фиг. 19-21).
После проведения вышеописанной процедуры было обнаружено, что независимо от метода предварительной стабилизации области антигена F (содержащей аминокислотную замену для предварительной стабилизации), если пре-F иммобилизуют на каркасных частицах посредством домена, присоединенного к С-концу его аминокислотной последовательности, то образование тримера улучшается, и при этом, сохранается способность антитела связываться с эпитопом Φ. Кроме того, было показано, что величина, при которой обнаруживается эпитоп I, который, как известно, индуцирует антитело, обладающее довольно низкой способностью нейтрализовать вирусную инфекцию, снижается. Приведенный выше результат позволяет предположить, что если антигены в виде частиц используются в качестве вакцинных антигенов, то предпочтительно индуцируется группа антител, которые, как считается, обладают высокой способностью нейтрализовать вирус.
[0140]
Анализ на встраивание линкера
Способность F_DS-Cav1, F_DS-Cav1-4GS-VLP, F_DS-Cav1-2GS-VLP, F_DS-Cav1-1GS-VLP, F_DS-Cav1-VLP, FH_85, FH_85-4GS-VLP, FH_85-2GS-VLP, FH_85-1GS-VLP и FH_85-VLP, которые представляют собой антигены или антигены в виде частиц, связываться с антителами, оценивали с помощью процедуры, описанной в Примере 6 (Фиг. 22-24).
После проведения вышеописанной процедуры было обнаружено, что при встраивании любого из линкеров GS достигается эффект улучшения образования тримера при сохранении способности антитела связываться с эпитопом Φ и наблюдается снижение величины, при которой детектируется эпитоп I. Однако, было показано, что эффект снижения уровня обнаружения эпитопа I ослабляется, если между каркасными частицами и антигеном встроен слишком длинный линкер GS. В частности, какого-либо значимого влияния не наблюдалось при длине приблизительно GGGGSGGGGS (SEQ ID NO: 19). Однако, для более длинной последовательности GGGGSGGGGSGGGGSGGGGS (SEQ ID NO: 75) наблюдалось ослабление эффекта снижения уровня обнаружения эпитопа I.
Приведенный выше результат свидетельствует о том, что, если гибкий линкер, имеющий определенную длину или более, встроен между каркасными частицами и антигеном, то эффект снижения доступности пост-F-распознающего антитела к сайту распознавания ослабляется за счет эффекта стерических затруднений.
[0141]
Оценка плотности антигена
Теоретический диаметр частиц антигена FH_85-VLP был оценен с применением метода динамического рассеяния света с использованием Zetasizer μV (Malvern), и составлял 89,28 нм (Фиг. 25). Поскольку в среднем присутствует приблизительно 135 молекул (приблизительно 45 молекул в виде тримера) на одну частицу, то средняя молекулярная плотность на 1 нм2 теоретической площади поверхности может составлять приблизительно 0,005 молекулы (приблизительно 0,0018 молекул в виде тримера). В данном случае, площадь поверхности (S) сферы вычисляли по следующей формуле с использованием ее радиуса (r): S=4πr2
[0142]
То есть, предполагается, что желаемый эффект стерических затруднений может быть достигнут в том случае, если плотность составляет по меньшей мере приблизительно 0,005 молекул антигена или более на 1 нм2 теоретической площади поверхности, оцененной с применением метода динамического рассеяния света.
Такой же результат был также получен для DS-Cav1-VLP и FH_82-VLP (фиг. 25).
[0143]
Подтверждение антител, индуцированных мышиным иммунитетом
Из приведенного выше эксперимента следует, что антиген пре-F, связанный с каркасными частицами HBsAg VLP, оказывает эффект подавления индуцирования иммунитета, который не способствует нейтрализации вируса. Для дальнейшего более подробного изучения такого эффекта был проведен анализ на антитела, индуцированные путем иммунизации мышей антигенами F_DS-Cav1, F_DS-Cav1-VLP, FH_82, FH_82-VLP, FH_85 или FH_85-VLP.
Иммунные сыворотки мышей получали путем иммунизации мышей BALB/c (самок в возрасте 5-8 недель) антигенами в виде частиц. В качестве композиции растворителя и каждого из антигенов в виде частиц использовали тщательно перемешанный раствор PBS или PBS с гидроксидом алюминия (InvivoGen, vac-alu-250) в равных количествах. Антиген вводили мышам внутримышечно в задние отделы бедра дважды с интервалом приблизительно в 3 недели и брали кровь приблизительно через 3 недели после второго введения. Полученную кровь инкубировали при комнатной температуре, а затем центрифугировали для сбора сыворотки.
ELISA-анализ, в котором антиген каждой конформации был иммобилизован, проводили для того, чтобы подтвердить степень индуцирования антитела, которое связывается с пре-F или пост-F в сыворотке.
Кроме того, для вычисления скорости индуцирования антитела, распознающего пост-F, проводили процедуру адсорбции антигеном и измеряли скорость уменьшения уровня антитела, распознающего антиген, посредством проведения процедуры адсорбции антигеном. Этот способ соответствует способу, описанному в Примере 5.
В заключение следует отметить, что благодаря образованию частиц подтверждается эффект предпочтительного индуцирования группы антител, которые, как считается, обладают высокой способностью нейтрализовать вирус.
[0144]
[Пример 8] Образование частиц антигена с использованием гидрофобной пептидной цепи
Была рассмотрена возможность того, что эффект снижения доступности антител или различных иммунных клеток к сайту антигена, распознаваемому антителом, распознающим пост-F, как показано в Примере 7, за счет эффекта стерического затруднения, также может быть достигнут с применением метода, в котором не используются каркасные частицы. Поэтому, в настоящем Примере описана оценка образования частиц путем гидрофобной агрегации с использованием гидрофобной пептидной цепи (полипептидной цепи, содержащей гидрофобную аминокислоту).
[0145]
Оценка гидрофобной пептидной цепи
Используемая гидрофобная пептидная цепь должна обладать способностью к гидрофобной агрегации в вакцинном препарате или в условиях in vivo, но ее трудно применять на практике для последовательности, которая значительно снижает эффективность секреторной экспрессии антигена клетками. В целом известно, что при слиянии с белком полипептидной цепи, содержащей большое количество гидрофобных аминокислот, эффективность продуцирования белка в клетках снижается. Поэтому были исследованы подходящие гидрофобные полипептидные цепи.
Антигены получали на основе белков-предшественников, в которых, последовательность (SEQ ID NO: 22), содержащая последовательность распознавания тромбином, His-метку и Strep-метку II в белке-предшественнике антигена F_DS-Cav1, была заменена последовательностями (SEQ ID NO: 44-47), которые содержат различные гидрофобные пептидные цепи (SEQ ID NO: 40-43) и метку FLAG, которая является меткой для очистки. Исследованные последовательности гидрофобных пептидов систематизированы в Таблице 8-1.
[0146]
Полинуклеотид, кодирующий каждый из вышеуказанных белков-предшественников, встраивали в вектор pcDNA3.4 (Thermo Fisher Scientific, Inc.) с применением стандартного метода генной инженерии. Встраивание последовательности ДНК-мишени было подтверждено с помощью анализа последовательности оснований, и был сконструирован каждый из экспрессионных векторов клеток млекопитающих, экспрессирующих белки-предшественники антигенов. Вектор для введения в клетку очищали с применением стандартного метода очистки плазмидного вектора.
[0147]
Экспрессию каждого из антигенов проводили в соответствии с процедурой, описанной в Примере 7.
Очистку каждого антигена проводили путем аффинной очистки с использованием аффинного геля, содержащего антитело против FLAG M2 (Sigma, A2220). Пептид FLAG использовали для элюирования при аффинной очистке. Однако, пептид FLAG в каждом образце антигена удаляли с помощью ультрафильтрационной мембраны. В качестве растворителя использовали PBS (pH 7,4).
Возможность экспрессии секретируемого антигена определяли путем проведения общего электрофореза в ДСН-ПААГ после очистки и подтверждения возможности выделения и очистки белка-мишени с высокой степенью чистоты. Кроме того, возможность образования частиц была подтверждена путем проведения обычного электрофореза в нативном ПААГ с добавлением синего красителя после очистки.
[0148]
Таблица 8-1
[0149]
Были изучены возможности экспрессии секретируемого антигена и образования частиц F_DS-Cav1-CC02 и F_DS-Cav1-CC03, и было обнаружено, что F_DS-Cav1-CC03 секретируется и экспрессируется, но не образует частицы, а F_DS-Cav1-CC02 недостаточно секретируется и экспрессируется (Фиг. 26). Поэтому считается, что оптимальная гидрофобность используемой гидрофобной пептидной цепи находится в пределах между гидрофобностями этих двух антигенов.
Различие между гидрофобными пептидными цепями F_DS-Cav1-CC02 и F_DS-Cav1-CC03 заключается в том, что аланин, который является негидрофильной аминокислотой, заменен лизином или глутаминовой кислотой, которая является гидрофильной аминокислотой. Поэтому сначала было принято решение заменить аланин F_DS-Cav1-CC02 на гистидин, обладающий умеренной гидрофильностью и имеющий рК, соответствующий приблизительно pH 6. Поскольку известно, что pH растворителя при приготовлении фармацевтического препарата является близким к нейтральному, а pH в секреторном пути клеток является более низким, то ожидалось, что пептидная цепь, содержащая гистидин, будет обладать относительно гидрофильным свойством во время экспрессии секретируемого антигена и относительно гидрофобным свойством во время приготовления фармацевтической композиции.
Кроме того, если учесть, что сконструированная гидрофобная пептидная цепь образует суперспирализованную структуру, то ожидается, что фрагменты гидрофобной пептидной цепи из 21 аминокислоты, соединенные непосредственно в присутствии фолдонового домена, представляют собой defgabcdefgabcdefgabc в указанном порядке. Если лейцин и изолейцин располагаются в положениях (a) и (d), которые гидрофобно взаимодействуют в тримере, согласно предыдущему исследованию (Joel P. Schneider et al., 1998, Folding & Design, 3:R29), в котором оценивали аминокислотные последовательности природных белков, то поскольку изолейцин в положении (а) и лейцин в положении (d) встречаются чаще, то считается, что подходящей заменой является замена лейцина и изолейцина в положении (а) и в положении (d) F_DS-Cav1-CC02.
Исходя из вышеизложенного, F_DS-Cav1-CC07 был заново сконструирован, и была исследована возможность экспрессии секретируемого антигена и возможность образования частиц, и были достигнуты как экспрессия секретируемого антигена, так и образование частиц (Фиг. 27). Поскольку экспрессия секретируемого антигена затруднена для F_DS-Cav1-CC08, в котором заменены положения гистидина, лизина и глутаминовой кислоты, также было высказано предположение, что проблема заключается не только в составе аминокислот, а также в достижении трехмерной реаранжировки.
[Химическая формула 1]
(взята из Folding & Design 1998, 3:R29)
[0150]
(Приготовление антигена в виде частиц)
Антигены получали на основе белков-предшественников (SEQ ID NO: 48 и 49), в которых последовательность (SEQ ID NO: 22), содержащая последовательность распознавания тромбином, His-метку и Strep-метку II в белках-предшественниках антигенов FH_82 и FH_85, была заменена последовательностью, которая содержит оптимальную гидрофобную пептидную цепь (SEQ ID NO: 42) и метку FLAG, которая является меткой для очистки.
[0151]
Полинуклеотид, кодирующий вышеуказанные белки-предшественники, встраивали в вектор pcDNA3.4 (Thermo Fisher Scientific, Inc.) с применением стандартного метода генной инженерии. Встраивание последовательности ДНК-мишени было подтверждено с помощью анализа последовательности оснований, и был сконструирован каждый из экспрессионных векторов клеток млекопитающих, экспрессирующих белки-предшественники. Вектор для введения в клетку очищали с применением стандартного метода очистки плазмидного вектора.
[0152]
Экспрессию каждого антигена осуществляли в соответствии с процедурой, описанной в анализе гидрофобных пептидных цепей.
Очистку каждого антигена проводили в соответствии с процедурой, описанной в анализе гидрофобных пептидных цепей.
[0153]
Антигены (которые также могут называться «антигенами в виде частиц»), используемые в анализе, описанном в этом Примере, систематизированы в Таблице 8-2.
[0154]
Таблица 8-2
[0155]
Анализ на способность связываться с антителом
Способность полученных антигенов в виде частиц связываться с антителами оценивали в соответствии с процедурой, описанной в Примере 6 (фиг. 28-30).
После проведения вышеописанной процедуры было обнаружено, что независимо от метода предварительной стабилизации области антигена F (содержащей аминокислотную замену для предварительной стабилизации), антиген в виде частиц, агрегированный гидрофобной пептидной цепью, имеющей пре-F, добавленный к С-концу его аминокислотной последовательности, улучшает образование тримера, сохраняя при этом способность антитела связываться с эпитопом Φ. Кроме того, был получен результат, показывающий, что обнаружение эпитопа I, который, как известно, индуцирует антитело, обладающее довольно низкой способностью нейтрализовать вирусную инфекцию, является сниженным. Приведенные выше результаты позволяют предположить, что при использовании антигена в качестве вакцинного антигена за счет образования частиц улучшается эффект преимущественного индуцирования группы антител, которые, как считается, обладают высокой способностью нейтрализовать вирус.
[0156]
Оценка плотности антигена
Теоретические диаметры антигена FH_85 и антигена в виде частиц FH_85-CC07 были оценены с применением метода динамического рассеяния света с использованием планшет-ридера DynaPro Plate Reader III (Wyatt) и составляли 13,8 нм и 60,0 нм (Фиг. 31). Поскольку отношение молекулярных масс двух частиц было пропорционально отношению их теоретических диаметров во 2-й и 3-й степени для обычных белков, то предполагается, что молекулярная масса FH_85-CC07 приблизительно в 20-80 раз больше молекулярной массы FH_85. То есть, считается, что FH85-CC07 составляет приблизительно 60-240 молекул на частицу (приблизительно 20-80 молекул в виде тримера), а средняя молекулярная плотность на 1 нм2 теоретической площади поверхности может составлять приблизительно 0,005-0,022 молекул (приблизительно 0,0017-0,0073 молекулы в виде тримера).
Такие же результаты были получены для FH_82-CC07 (Фиг. 31).
Таким образом, было установлено, что антиген в виде частиц, полученный с использованием гидрофобной пептидной цепи, имеет плотность антигена, эквивалентную или превышающую плотность антигена в виде частиц, полученного с использованием каркасных частиц, представленных в Примере 6.
[0157]
Подтверждение антител, индуцированных мышиным иммунитетом
С помощью описанной выше процедуры было высказано предположение, что образование частиц антигена, с которым связана гидрофобная пептидная цепь, дает эффект подавления индуцирования иммунитета, который не способствует нейтрализации вируса. Для более детального изучения этого эффекта был проведен анализ антител, индуцированных путем иммунизации мышей антигеном или антигеном в виде частиц F_DS-Cav1F_DS-Cav1-CC07, FH_82-CC07 или FH_85-CC07.
[0158]
Мышиные иммунные сыворотки получали путем иммунизации BALB/cAnNCrlCrlj (Charles River Laboratories Japan) (самок в возрасте 7 недель) антигенами и антигенами в виде частиц. Антиген или антиген в виде частиц F_DS-Cav1, F_DS-Cav1-CC07, FH_82-CC07 или FH_85-CC07 (концентрация антигена: 0,40 мг/мл), растворенные в PBS (рН 7,4), вводили внутримышечно в количестве 50 мкл в задний отдел бедра мышей на 0-й день иммунизации и на 21-й день иммунизации (по 5 мышей в группе). Кровь брали из задней полой вены на 42-й день иммунизации, и полученную кровь инкубировали (при комнатной температуре, приблизительно 2 часа, в стационарных условиях), а затем сыворотку собирали путем центрифугирования и инактивировали (при 55°С, 30 минут) для приготовления мышиной иммунной сыворотки.
[0159]
Для того, чтобы подтвердить соотношение, при котором индуцируются антитела, которые связываются с конформациями после слияния, с применением метода, описанного в Примере 6, каждую из мышиных иммунных сывороток подвергали адсорбции антигеном с использованием магнитных шариков, к которым присоединяли ΔFP furinwt Fecto, или магнитных шариков, которые были подвергнуты только процедуре блокирования без связывания какого-либо антигена. Кроме того, для каждой из сывороток после процедуры адсорбции проводили ELISA-анализ, в котором F_DS-Cav1 иммобилизовали с применением метода, описанного в Примере 6, и вычисляли скорость адсорбции. Однако, скорость адсорбции устанавливали равной относительному значению, полученному путем вычитания титра антител в сыворотке после процедуры адсорбции с использованием антигена ΔFP furinwt Fecto из титра антител в сыворотке после процедуры адсорбции но без антигена, если титр антител сыворотки после процедуры адсорбции без антигена был принят за 100% (Фиг. 32).
[0160]
После проведения вышеописанной процедуры было показано, что пост-F-распознающее антитело, которое приблизительно наполовину индуцировалось антигеном F_DS-Cav1 до образования частиц, почти не индуцировалось антигеном F_DS-Cav1-CC07, образующим частицы посредством гидрофобной пептидной цепи. Кроме того, аналогичным образом, антитело, распознающее пост-F, также почти не индуцировалось антигенами FH_82-CC07 и FH_85-CC07. Исходя из вышеизложенного, предполагается, что за счет образования частиц улучшается эффект преимущественного индуцирования группы антител, которые, как считается, обладают высокой способностью нейтрализовать вирус.
[0161]
[Пример 9] Образование частиц антигена с использованием самоассоциирующегося белкового домена
Рассматривалась возможность того, что эффект снижения доступности антител или различных иммунных клеток к антигенному сайту, распознаваемому пост-F-распознающим антителом, как показано в Примерах 7 и 8, за счет эффекта стерических затруднений, также может быть реализован путем слияния самоассоциирующегося белкового домена с С-концевой стороной антигена. В частности, благодаря регулярному расположению антигенов с использованием самоассоциирующегося белкового домена рассматривалась возможность достижения более высокого эффекта стерических затруднений, чем образование частиц путем гидрофобной агрегации, как показано в Примере 8.
Таким образом, в этом Примере исследовали образование частиц с использованием корового антигена (HBcAg) вируса гепатита В, обладающего способностью к самоассоциации.
Однако, в тех случаях, когда была проведена замена последовательности (SEQ ID NO: 22), содержащей последовательность распознавания тромбином, His-метку и Strep-метку II в антигене F_DS-Cav1, на пептидную последовательность (SEQ ID NO: 51), которая содержит линкер GS (GGGGSGGGGSGGGGSGGGGS) (SEQ ID NO: 75), самоассоциирующийся домен HBcAg (SEQ ID NO: 50) и His-метку, то, несмотря на наблюдаемую экспрессию, самоассоциация не обнаруживалась (результат такого анализа был подтвержден с помощью электрофореза в нативном ПААГ с окрашиванием синим красителем). N-конец самоассоциирующегося домена HBcAg присутствует рядом с поверхностью связывания, если HbcAg подвергается самоассоциации, и рассматривалась возможность того, что самоассоциация может быть затруднена в результате слияния крупного белка с N-концом.
Таким образом, чтобы расположить F-антигенный фрагмент RSV в положении, удаленном от поверхности связывания во время самоассоциации, если HBcAg подвергается такой самоассоции, то его нужно разделить на С-концевую сторону и N-концевую сторону, но не на выступающую наружу сторону частицы (фрагмент между спиралями, присутствующими в домене α3 и домене α4). Кроме того, чтобы снизить риск неожиданного образования дисульфидной связи, были введены две точковые мутации (C48S и C107A), которые, как предполагается, не влияют на самоассоциацию. В частности, антигены (которые также могут называться «антигенами в виде частиц») получали на основе белка-предшественника (SEQ ID NO: 55) F_DS-Cav1-HBcCN и белка-предшественника (SEQ ID NO: 56) FH_85-HBcCN, в каждом из которых последовательность (SEQ ID NO: 22), содержащая последовательность распознавания тромбином, His-метку и Strep-метку II в белках-предшественниках антигенов F_DS-Cav1 и FH_85, была заменена последовательностью, содержащей линкер GS (GGGGSGGGGSGGGGSGGGGS) (SEQ ID NO: 75), С-концевую сторону самоассоциирующегося домена HBcAg (SEQ ID NO: 52) и последовательность (SEQ ID NO: 54), в которой линкер GS (GGGGSGGGGSGGGGSGGGGS) (SEQ ID NO: 20), N-концевая сторона самоассоциирующегося домена HBcAg (SEQ ID NO: 53) и метка FLAG связаны в указанном порядке.
F_DS-Cav1-HBcCN экспрессировали и очищали с помощью процедуры, описанной в Примере 8, и исследовали возможность экспрессии секретируемого антигена и возможность образования частиц, в результате чего была достигнута как экспрессия секретируемого антигена, так и образование частиц (фиг. 33). Однако, поскольку относительный уровень экспрессии был низким, то было бы желательно усовершенствование метода экспрессии. Таким образом, для полинуклеотида, кодирующего предшественник DS-Cav1-HBcCN или FH_85-HBcCN, была создана линия CHO со стабильной экспрессией с использованием системы экспрессии генов GS Xceed (Lonza). Полученную клеточную линию культивировали, и после секреции и экспрессии каждого антигена получали компонент культурального супернатанта.
Очистку каждого антигена в виде частиц осуществляли с помощью процедуры, описанной в Примере 8.
Для подтверждения способности каждого антигена в виде частиц связываться с каждым антителом был проведен ELISA-анализ, в котором были иммобилизованы паливизумаб (внутримышечная инъекция Synagis, AbbVie, Inc.), антитело 131-2A и антитело D25. Карбонатно-бикарбонатный буфер (Sigma-Aldrich, C3041-50CAP) использовали в качестве буфера для связывания антител, а TBS (TaKaRa, T9142) использовали для разведения образца и промывки планшета. 10 мкг/мл паливизумаба, или антитела 131-2A, или антитела D25 добавляли в половину лунок 96-луночного планшета для прямой иммобилизации и блокирования альбумином бычьей сыворотки. Серию 10-кратных разведений приготавливали путем соответствующего добавления буфера к каждому антигену и добавляли в планшет для инкубирования (при комнатной температуре, 2 часа, в стационарных условиях). После этого, добавленный раствор антигена удаляли и подвергали связыванию с конъюгированным с пероксидазой антителом против FLAG М2 (Sigma-Aldrich, A8592), а затем проявляли окраску с помощью стандартного реагента для обнаружения пероксидазы, после чего, интенсивность проявления окраски определяли путем измерения поглощения на стандартном планшет-ридере. Зависимость между скоростью разведения антигена и поглощением вычисляли путем кусочно-линейной интерполяции между точками измерения, и обратная величина степени разведения, при котором поглощение равно 0,2, была использована как сила связывания добавленного антигена с иммобилизованным антителом. Значение, полученное путем деления силы связывания с антителом D25 на силу связывания с паливизумабом, определяли как сигнал обнаружения эпитопа Φ, а значение, полученное путем деления силы связывания с антителом 131-2A, на силу связывания с паливизумабом, определяли как сигнал обнаружения эпитопа I (фиг. 34 и 35).
С помощью описанной выше процедуры был получен результат, показывающий, что по сравнению с образованием частиц посредством гидрофобной пептидной цепи, антиген в виде частиц, агрегированных под действием домена самоассоциирующегося белка с пре-F, добавленного к С-концу аминокислотной последовательности, также заметно улучшает способность антитела связываться с эпитопом Φ и снижает степень обнаружения эпитопа I, который, как известно, индуцирует антитело, обладающее довольно низкой способностью нейтрализовать вирусную инфекцию. Приведенный выше результат свидетельствует о том, что за счет образования частиц улучшается эффект преимущественного индуцирования группы антител, которые, как считается, обладают высокой активностью в отношении нейтрализации вируса.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Mitsubishi Tanabe Pharma Corporation
<120> Мутантный белок F RSV и его применение
<130> 19084-201016
<150> US62/952673
<151> 2019-12-23
<160> 75
<170> PatentIn version 3.5
<210> 1
<211> 574
<212> PRT
<213> Респираторно-синцитиальный вирус
<220>
<221> MISC_FEATURE
<222> (1)..(25)
<223> сигнальный пептид
<220>
<221> MISC_FEATURE
<222> (26)..(109)
<223> F2
<220>
<221> MISC_FEATURE
<222> (110)..(136)
<223> pep27
<220>
<221> MISC_FEATURE
<222> (137)..(513)
<223> внеклеточный F1
<220>
<221> MISC_FEATURE
<222> (514)..(574)
<223> трансмембранный и внутриклеточный F1
<400> 1
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Pro Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Ser Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Ser Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Val Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Ser Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Ile Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Met Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu His Asn Val Asn Ala Gly Lys Ser Thr Thr Asn Ile Met Ile Thr
515 520 525
Thr Ile Ile Ile Val Ile Ile Val Ile Leu Leu Ser Leu Ile Ala Val
530 535 540
Gly Leu Leu Leu Tyr Cys Lys Ala Arg Ser Thr Pro Val Thr Leu Ser
545 550 555 560
Lys Asp Gln Leu Ser Gly Ile Asn Asn Ile Ala Phe Ser Asn
565 570
<210> 2
<211> 461
<212> PRT
<213> Респираторно-синцитиальный вирус
<220>
<221> MISC_FEATURE
<222> (1)..(84)
<223> F2
<220>
<221> MISC_FEATURE
<222> (85)..(461)
<223> внеклеточный F1
<400> 2
Gln Asn Ile Thr Glu Glu Phe Tyr Gln Ser Thr Cys Ser Ala Val Ser
1 5 10 15
Lys Gly Tyr Leu Ser Ala Leu Arg Thr Gly Trp Tyr Thr Ser Val Ile
20 25 30
Thr Ile Glu Leu Ser Asn Ile Lys Glu Asn Lys Cys Asn Gly Thr Asp
35 40 45
Ala Lys Val Lys Leu Ile Lys Gln Glu Leu Asp Lys Tyr Lys Asn Ala
50 55 60
Val Thr Glu Leu Gln Leu Leu Met Gln Ser Thr Pro Pro Thr Asn Asn
65 70 75 80
Arg Ala Arg Arg Phe Leu Gly Phe Leu Leu Gly Val Gly Ser Ala Ile
85 90 95
Ala Ser Gly Val Ala Val Ser Lys Val Leu His Leu Glu Gly Glu Val
100 105 110
Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys Ala Val Val Ser
115 120 125
Leu Ser Asn Gly Val Ser Val Leu Thr Ser Lys Val Leu Asp Leu Lys
130 135 140
Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Val Asn Lys Gln Ser Cys
145 150 155 160
Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln Gln Lys Asn Asn
165 170 175
Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn Ala Gly Val Thr
180 185 190
Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu Leu Leu Ser Leu
195 200 205
Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys Leu Met Ser Asn
210 215 220
Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile Met Ser Ile Ile
225 230 235 240
Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro Leu Tyr Gly Val
245 250 255
Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro Leu Cys Thr Thr
260 265 270
Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg Thr Asp Arg Gly
275 280 285
Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe Pro Gln Ala Glu
290 295 300
Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp Thr Met Asn Ser
305 310 315 320
Leu Thr Leu Pro Ser Glu Ile Asn Leu Cys Asn Val Asp Ile Phe Asn
325 330 335
Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr Asp Val Ser Ser
340 345 350
Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys Tyr Gly Lys Thr
355 360 365
Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile Lys Thr Phe Ser
370 375 380
Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Met Asp Thr Val Ser Val
385 390 395 400
Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly Lys Ser Leu Tyr
405 410 415
Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro Leu Val Phe Pro
420 425 430
Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn Glu Lys Ile Asn
435 440 445
Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu Leu
450 455 460
<210> 3
<211> 488
<212> PRT
<213> респираторно-синцитиальный вирус
<220>
<221> MISC_FEATURE
<222> (1)..(84)
<223> F2
<220>
<221> MISC_FEATURE
<222> (85)..(111)
<223> pep27
<220>
<221> MISC_FEATURE
<222> (112)..(488)
<223> внеклеточный F1
<400> 3
Gln Asn Ile Thr Glu Glu Phe Tyr Gln Ser Thr Cys Ser Ala Val Ser
1 5 10 15
Lys Gly Tyr Leu Ser Ala Leu Arg Thr Gly Trp Tyr Thr Ser Val Ile
20 25 30
Thr Ile Glu Leu Ser Asn Ile Lys Glu Asn Lys Cys Asn Gly Thr Asp
35 40 45
Ala Lys Val Lys Leu Ile Lys Gln Glu Leu Asp Lys Tyr Lys Asn Ala
50 55 60
Val Thr Glu Leu Gln Leu Leu Met Gln Ser Thr Pro Pro Thr Asn Asn
65 70 75 80
Arg Ala Arg Arg Glu Leu Pro Arg Phe Met Asn Tyr Thr Leu Asn Asn
85 90 95
Ala Lys Lys Thr Asn Val Thr Leu Ser Lys Lys Arg Lys Arg Arg Phe
100 105 110
Leu Gly Phe Leu Leu Gly Val Gly Ser Ala Ile Ala Ser Gly Val Ala
115 120 125
Val Ser Lys Val Leu His Leu Glu Gly Glu Val Asn Lys Ile Lys Ser
130 135 140
Ala Leu Leu Ser Thr Asn Lys Ala Val Val Ser Leu Ser Asn Gly Val
145 150 155 160
Ser Val Leu Thr Ser Lys Val Leu Asp Leu Lys Asn Tyr Ile Asp Lys
165 170 175
Gln Leu Leu Pro Ile Val Asn Lys Gln Ser Cys Ser Ile Ser Asn Ile
180 185 190
Glu Thr Val Ile Glu Phe Gln Gln Lys Asn Asn Arg Leu Leu Glu Ile
195 200 205
Thr Arg Glu Phe Ser Val Asn Ala Gly Val Thr Thr Pro Val Ser Thr
210 215 220
Tyr Met Leu Thr Asn Ser Glu Leu Leu Ser Leu Ile Asn Asp Met Pro
225 230 235 240
Ile Thr Asn Asp Gln Lys Lys Leu Met Ser Asn Asn Val Gln Ile Val
245 250 255
Arg Gln Gln Ser Tyr Ser Ile Met Ser Ile Ile Lys Glu Glu Val Leu
260 265 270
Ala Tyr Val Val Gln Leu Pro Leu Tyr Gly Val Ile Asp Thr Pro Cys
275 280 285
Trp Lys Leu His Thr Ser Pro Leu Cys Thr Thr Asn Thr Lys Glu Gly
290 295 300
Ser Asn Ile Cys Leu Thr Arg Thr Asp Arg Gly Trp Tyr Cys Asp Asn
305 310 315 320
Ala Gly Ser Val Ser Phe Phe Pro Gln Ala Glu Thr Cys Lys Val Gln
325 330 335
Ser Asn Arg Val Phe Cys Asp Thr Met Asn Ser Leu Thr Leu Pro Ser
340 345 350
Glu Ile Asn Leu Cys Asn Val Asp Ile Phe Asn Pro Lys Tyr Asp Cys
355 360 365
Lys Ile Met Thr Ser Lys Thr Asp Val Ser Ser Ser Val Ile Thr Ser
370 375 380
Leu Gly Ala Ile Val Ser Cys Tyr Gly Lys Thr Lys Cys Thr Ala Ser
385 390 395 400
Asn Lys Asn Arg Gly Ile Ile Lys Thr Phe Ser Asn Gly Cys Asp Tyr
405 410 415
Val Ser Asn Lys Gly Met Asp Thr Val Ser Val Gly Asn Thr Leu Tyr
420 425 430
Tyr Val Asn Lys Gln Glu Gly Lys Ser Leu Tyr Val Lys Gly Glu Pro
435 440 445
Ile Ile Asn Phe Tyr Asp Pro Leu Val Phe Pro Ser Asp Glu Phe Asp
450 455 460
Ala Ser Ile Ser Gln Val Asn Glu Lys Ile Asn Gln Ser Leu Ala Phe
465 470 475 480
Ile Arg Lys Ser Asp Glu Leu Leu
485
<210> 4
<211> 562
<212> PRT
<213> Искусственная последовательность
<220>
<223> нативный F
<220>
<221> MISC_FEATURE
<222> (1)..(25)
<223> сигнальный пептид
<220>
<221> MISC_FEATURE
<222> (26)..(109)
<223> F2
<220>
<221> MISC_FEATURE
<222> (110)..(136)
<223> pep27
<220>
<221> MISC_FEATURE
<222> (137)..(513)
<223> внеклеточный F1
<220>
<221> MISC_FEATURE
<222> (514)..(542)
<223> фолдон
<220>
<221> MISC_FEATURE
<222> (543)..(562)
<223> метка
<400> 4
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Ser Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Ser Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Val Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Ser Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Leu Val
530 535 540
Pro Arg Gly Ser His His His His His His Trp Ser His Pro Gln Phe
545 550 555 560
Glu Lys
<210> 5
<211> 1686
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нативный F
<400> 5
atggaactcc tcattcttaa ggctaacgcc ataaccacga ttctgactgc tgtgaccttc 60
tgctttgcta gcgggcaaaa cattaccgaa gagttctatc aatccacttg tagcgcggtt 120
agtaagggct atctatccgc cttaaggact ggctggtaca cttccgtcat tacgattgag 180
ctgagtaata tcaaggagaa caaatgtaac ggcacagatg ccaaggtgaa actgattaag 240
caggagcttg acaagtacaa gaatgccgtt accgagctgc agttgctgat gcagagcaca 300
ccagcgacca ataatcgggc aagacgcgaa cttcctcggt ttatgaacta taccttgaat 360
aatgctaaga agacaaacgt cacactgtca aagaaacgaa aacgtaggtt cctcggcttc 420
cttctcggag taggcagtgc catcgcaagt ggagtagcag tctccaaagt gttgcaccta 480
gagggagaag tgaacaagat caaatctgca ctcctgagca ccaacaaagc tgtcgtctct 540
ttatccaatg gcgtttcagt gctcacgtct aaggttctcg acctgaaaaa ctacatcgac 600
aaacagctcc ttcccatcgt caacaaacag agttgctcga tttctaacat cgagacagtt 660
atcgaattcc aacagaagaa caatagactt ctggagatca ctcgggagtt ttccgtaaat 720
gcaggtgtga ctactccggt ctcaacctac atgctgacta attccgagtt attgtcgcta 780
atcaacgata tgcctataac gaatgaccag aagaaactca tgagcaacaa cgtgcagatc 840
gtaaggcagc aatcctactc catcatgtcc ataatcaagg aagaggtgct ggcgtatgta 900
gtccagttgc cactgtatgg agtgatagac accccatgtt ggaagctcca tacgagcccc 960
ctgtgtacca ctaatacaaa ggaggggtct aacatatgcc ttacccggac tgatcgtggg 1020
tggtattgcg acaatgccgg ttcagtgtcg ttctttccac aagccgaaac atgtaaggtg 1080
cagtcaaacc gagtgttctg tgacacaatg aatagcttga cattgccctc tgaggtgaac 1140
ctgtgcaatg tggacatttt caaccccaag tacgactgca aaatcatgac aagcaaaact 1200
gacgtgtcta gctctgtcat tacctctcta ggagccattg tgagctgcta cggtaagaca 1260
aaatgcacag cttcaaacaa gaatagaggc atcatcaaga ccttctccaa tgggtgtgat 1320
tacgtgagca ataaaggtgt ggacaccgtt agcgtaggca ataccctgta ttacgttaat 1380
aagcaggaag gcaaaagtct gtacgtcaaa ggggagccca tcataaactt ctacgatcct 1440
ctcgtgtttc caagcgatga gtttgatgcc tccatttcac aggtgaacga aaagatcaac 1500
cagtctctgg cctttattcg caaaagtgat gaactgctgg gaagcgggta tattcccgaa 1560
gctcctaggg atggacaagc atatgtgcgc aaggatggtg aatgggtcct gctgtctacc 1620
tttctgttag ttccgagagg gagtcatcat caccaccatc actggtcaca ccctcagttt 1680
gagaaa 1686
<210> 6
<211> 488
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_50
<220>
<221> MISC_FEATURE
<222> (1)..(84)
<223> F2
<220>
<221> MISC_FEATURE
<222> (85)..(111)
<223> pep27
<220>
<221> MISC_FEATURE
<222> (112)..(488)
<223> внеклеточный F1
<400> 6
Gln Asn Ile Thr Glu Glu Phe Tyr Gln Ser Thr Cys Ser Ala Val Ser
1 5 10 15
Lys Gly Tyr Leu Ser Ala Leu Arg Thr Gly Trp Tyr Thr Ser Val Ile
20 25 30
Thr Ile Glu Leu Ser Asn Ile Lys Glu Asn Lys Cys Asn Gly Thr Asp
35 40 45
Ala Lys Val Lys Leu Ile Lys Gln Glu Leu Asp Lys Tyr Lys Asn Ala
50 55 60
Val Thr Glu Leu Gln Leu Leu Met Gln Ser Thr Pro Ala Thr Asn Asn
65 70 75 80
Arg Ala Arg Arg Glu Leu Pro Arg Phe Met Asn Tyr Thr Leu Asn Asn
85 90 95
Ala Lys Lys Thr Asn Val Thr Leu Ser Lys Lys Arg Lys Asn Asn Phe
100 105 110
Leu Gly Phe Cys Leu Gly Val Gly Ser Ala Ile Ala Ser Gly Val Ala
115 120 125
Val Ser Lys Val Leu His Leu Glu Gly Glu Val Asn Lys Ile Lys Ser
130 135 140
Ala Leu Leu Ser Thr Asn Lys Ala Val Val Ser Leu Ser Asn Gly Val
145 150 155 160
Ser Val Leu Val Val Lys Val Leu Asp Leu Lys Asn Tyr Ile Asp Lys
165 170 175
Gln Leu Leu Pro Ile Val Asn Lys Gln Ser Cys Ser Ile Ser Asn Ile
180 185 190
Glu Thr Val Ile Glu Phe Gln Gln Lys Asn Asn Arg Leu Leu Glu Ile
195 200 205
Thr Arg Glu Phe Ser Val Asn Ala Gly Val Thr Thr Pro Val Ser Thr
210 215 220
Tyr Met Leu Thr Asn Ser Glu Leu Leu Ser Leu Ile Asn Asp Met Pro
225 230 235 240
Ile Thr Asn Asp Gln Lys Lys Leu Met Ser Asn Asn Val Gln Ile Val
245 250 255
Arg Gln Gln Ser Tyr Ser Ile Met Ser Ile Ile Lys Glu Glu Val Leu
260 265 270
Ala Tyr Val Val Gln Leu Pro Leu Tyr Gly Val Ile Asp Thr Pro Cys
275 280 285
Trp Lys Leu His Thr Ser Pro Leu Cys Thr Thr Asn Thr Lys Glu Gly
290 295 300
Ser Asn Ile Cys Leu Thr Arg Thr Asp Arg Gly Trp Tyr Cys Asp Asn
305 310 315 320
Ala Gly Ser Val Ser Phe Phe Pro Gln Ala Glu Thr Cys Lys Val Gln
325 330 335
Ser Asn Arg Val Phe Cys Asp Thr Met Asn Ser Cys Thr Leu Pro Ser
340 345 350
Glu Val Asn Leu Cys Asn Val Asp Ile Phe Asn Pro Lys Tyr Asp Cys
355 360 365
Lys Ile Met Thr Ser Lys Thr Asp Val Ser Ser Ser Val Ile Thr Ser
370 375 380
Leu Gly Ala Ile Val Ser Cys Tyr Gly Lys Thr Lys Cys Thr Ala Ser
385 390 395 400
Asn Lys Asn Arg Gly Ile Ile Lys Thr Phe Ser Asn Gly Cys Asp Tyr
405 410 415
Val Ser Asn Lys Gly Val Asp Thr Val Ser Val Gly Asn Thr Leu Tyr
420 425 430
Tyr Val Asn Lys Gln Glu Gly Lys Ser Leu Tyr Val Lys Gly Glu Pro
435 440 445
Ile Ile Asn Phe Tyr Asp Pro Leu Val Phe Pro Ser Asp Glu Phe Asp
450 455 460
Ala Ser Ile Ser Gln Val Asn Glu Lys Ile Asn Gln Ser Leu Ala Phe
465 470 475 480
Ile Arg Lys Ser Asp Glu Leu Leu
485
<210> 7
<211> 488
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_81
<220>
<221> MISC_FEATURE
<222> (1)..(84)
<223> F2
<220>
<221> MISC_FEATURE
<222> (85)..(111)
<223> pep27
<220>
<221> MISC_FEATURE
<222> (112)..(488)
<223> внеклеточный F1
<400> 7
Gln Asn Ile Thr Glu Glu Phe Tyr Gln Ser Thr Cys Ser Ala Val Ser
1 5 10 15
Lys Gly Tyr Leu Ser Ala Leu Arg Thr Gly Trp Tyr Thr Ser Val Ile
20 25 30
Thr Ile Met Leu Ser Asn Ile Lys Glu Asn Lys Cys Asn Gly Thr Asp
35 40 45
Ala Lys Val Lys Leu Ile Lys Gln Glu Leu Asp Lys Tyr Lys Asn Ala
50 55 60
Val Thr Glu Leu Gln Leu Leu Met Gln Ser Thr Pro Ala Thr Asn Asn
65 70 75 80
Arg Ala Arg Arg Glu Leu Pro Arg Phe Met Asn Tyr Thr Leu Asn Asn
85 90 95
Ala Lys Lys Thr Asn Val Thr Leu Ser Lys Lys Arg Lys Asn Asn Phe
100 105 110
Leu Gly Phe Cys Leu Gly Val Gly Ser Ala Ile Ala Ser Gly Val Ala
115 120 125
Val Ser Lys Val Leu His Leu Glu Gly Glu Val Asn Lys Ile Lys Ser
130 135 140
Ala Leu Leu Ser Thr Asn Lys Ala Val Val Ser Leu Ser Asn Gly Val
145 150 155 160
Ser Val Leu Val Val Lys Val Leu Asp Leu Lys Asn Tyr Ile Asp Lys
165 170 175
Gln Leu Leu Pro Ile Val Asn Lys Gln Ser Cys Ser Ile Ser Asn Ile
180 185 190
Glu Thr Val Ile Glu Phe Gln Gln Lys Asn Asn Arg Leu Leu Glu Ile
195 200 205
Thr Arg Glu Phe Ser Val Asn Ala Gly Val Thr Thr Pro Val Ser Thr
210 215 220
Tyr Met Leu Thr Asn Ser Glu Leu Leu Ser Leu Ile Asn Asp Met Pro
225 230 235 240
Ile Thr Asn Asp Gln Lys Lys Leu Met Ser Asn Asn Val Gln Ile Val
245 250 255
Arg Gln Gln Ser Tyr Ser Ile Met Ser Ile Ile Lys Glu Glu Val Leu
260 265 270
Ala Tyr Val Val Gln Leu Pro Leu Tyr Gly Val Ile Asp Thr Pro Cys
275 280 285
Trp Lys Leu His Thr Ser Pro Leu Cys Thr Thr Asn Thr Lys Glu Gly
290 295 300
Ser Asn Ile Cys Leu Thr Arg Thr Asp Arg Gly Trp Tyr Cys Asp Asn
305 310 315 320
Ala Gly Ser Val Ser Phe Phe Pro Gln Ala Glu Thr Cys Lys Val Gln
325 330 335
Ser Asn Arg Val Phe Cys Asp Thr Met Asn Ser Cys Thr Leu Pro Ser
340 345 350
Glu Val Asn Leu Cys Asn Val Asp Ile Phe Asn Pro Lys Tyr Asp Cys
355 360 365
Lys Ile Met Thr Ser Lys Thr Asp Val Ser Ser Ser Val Ile Thr Ser
370 375 380
Leu Gly Ala Ile Val Ser Cys Tyr Gly Lys Thr Lys Cys Thr Ala Ser
385 390 395 400
Asn Lys Asn Arg Gly Ile Ile Lys Thr Phe Ser Asn Gly Cys Asp Tyr
405 410 415
Val Ser Asn Lys Gly Val Asp Thr Val Ser Val Gly Asn Thr Leu Tyr
420 425 430
Tyr Val Asn Lys Gln Glu Gly Lys Ser Leu Tyr Val Lys Gly Glu Pro
435 440 445
Ile Ile Asn Phe Tyr Asp Pro Leu Val Phe Pro Ser Asp Glu Phe Asp
450 455 460
Ala Ser Ile Ser Gln Val Asn Glu Lys Ile Asn Gln Ser Leu Ala Phe
465 470 475 480
Ile Arg Lys Ser Asp Glu Leu Leu
485
<210> 8
<211> 488
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_82
<220>
<221> MISC_FEATURE
<222> (1)..(84)
<223> F2
<220>
<221> MISC_FEATURE
<222> (85)..(111)
<223> pep27
<220>
<221> MISC_FEATURE
<222> (112)..(488)
<223> внеклеточный F1
<400> 8
Gln Asn Ile Thr Glu Glu Phe Tyr Gln Ser Thr Cys Ser Ala Val Ser
1 5 10 15
Arg Gly Tyr Leu Ser Ala Leu Arg Thr Gly Trp Tyr Thr Ser Val Ile
20 25 30
Thr Ile Glu Leu Ser Asn Ile Lys Glu Asn Lys Cys Asn Gly Thr Asp
35 40 45
Ala Lys Val Lys Leu Ile Lys Gln Glu Leu Asp Lys Tyr Lys Asn Ala
50 55 60
Val Thr Glu Leu Gln Leu Leu Met Gln Ser Thr Pro Ala Thr Asn Asn
65 70 75 80
Arg Ala Arg Arg Glu Leu Pro Arg Phe Met Asn Tyr Thr Leu Asn Asn
85 90 95
Ala Lys Lys Thr Asn Val Thr Leu Ser Lys Lys Arg Lys Asn Asn Phe
100 105 110
Leu Gly Phe Cys Leu Gly Val Gly Ser Ala Ile Ala Ser Gly Val Ala
115 120 125
Val Ser Lys Val Leu His Leu Glu Gly Glu Val Asn Lys Ile Lys Ser
130 135 140
Ala Leu Leu Ser Thr Asn Lys Ala Val Val Ser Leu Ser Asn Gly Val
145 150 155 160
Ser Val Leu Val Val Lys Val Leu Asp Leu Lys Asn Tyr Ile Asp Lys
165 170 175
Gln Leu Leu Pro Ile Val Asn Lys Gln Ser Cys Ser Ile Ser Asn Ile
180 185 190
Glu Thr Val Ile Glu Phe Gln Gln Lys Asn Asn Arg Leu Leu Glu Ile
195 200 205
Thr Arg Glu Phe Ser Val Asn Ala Gly Val Thr Thr Pro Val Ser Thr
210 215 220
Tyr Met Leu Thr Asn Ser Glu Leu Leu Ser Leu Ile Asn Asp Met Pro
225 230 235 240
Ile Thr Asn Asp Gln Lys Lys Leu Met Ser Asn Asn Val Gln Ile Val
245 250 255
Arg Gln Gln Ser Tyr Ser Ile Met Ser Ile Ile Lys Glu Glu Val Leu
260 265 270
Ala Tyr Val Val Gln Leu Pro Leu Tyr Gly Val Ile Asp Thr Pro Cys
275 280 285
Trp Lys Leu His Thr Ser Pro Leu Cys Thr Thr Asn Thr Lys Glu Gly
290 295 300
Ser Asn Ile Cys Leu Thr Arg Thr Asp Arg Gly Trp Tyr Cys Asp Asn
305 310 315 320
Ala Gly Ser Val Ser Phe Phe Pro Gln Ala Glu Thr Cys Lys Val Gln
325 330 335
Ser Asn Arg Val Phe Cys Asp Thr Met Asn Ser Cys Thr Leu Pro Ser
340 345 350
Glu Val Asn Leu Cys Asn Thr Asp Ile Phe Asn Pro Lys Tyr Asp Cys
355 360 365
Lys Ile Met Thr Ser Lys Thr Asp Val Ser Ser Ser Val Ile Thr Ser
370 375 380
Leu Gly Ala Ile Val Ser Cys Tyr Gly Lys Thr Lys Cys Thr Ala Ser
385 390 395 400
Asn Lys Asn Arg Gly Ile Ile Lys Thr Phe Ser Asn Gly Cys Asp Tyr
405 410 415
Val Ser Asn Lys Gly Val Asp Thr Val Ser Val Gly Asn Thr Leu Tyr
420 425 430
Tyr Val Asn Lys Gln Glu Gly Lys Ser Leu Tyr Val Lys Gly Glu Pro
435 440 445
Ile Ile Asn Phe Tyr Asp Pro Leu Val Phe Pro Ser Asp Glu Phe Asp
450 455 460
Ala Ser Ile Ser Gln Val Asn Glu Lys Ile Asn Gln Ser Leu Ala Phe
465 470 475 480
Ile Arg Lys Ser Asp Glu Leu Leu
485
<210> 9
<211> 488
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_85
<220>
<221> MISC_FEATURE
<222> (1)..(84)
<223> F2
<220>
<221> MISC_FEATURE
<222> (85)..(111)
<223> pep27
<220>
<221> MISC_FEATURE
<222> (112)..(488)
<223> внеклеточный F1 r
<400> 9
Gln Asn Ile Thr Glu Glu Phe Tyr Gln Ser Thr Cys Ser Ala Val Ser
1 5 10 15
Arg Gly Tyr Leu Ser Ala Leu Arg Thr Gly Trp Tyr Thr Ser Val Ile
20 25 30
Thr Ile Met Leu Ser Asn Ile Lys Glu Asn Lys Cys Asn Gly Thr Asp
35 40 45
Ala Lys Val Lys Leu Ile Lys Gln Glu Leu Asp Lys Tyr Lys Asn Ala
50 55 60
Val Thr Glu Leu Gln Leu Leu Met Gln Ser Thr Pro Ala Thr Asn Asn
65 70 75 80
Arg Ala Arg Arg Glu Leu Pro Arg Phe Met Asn Tyr Thr Leu Asn Asn
85 90 95
Ala Lys Lys Thr Asn Val Thr Leu Ser Lys Lys Arg Lys Asn Asn Phe
100 105 110
Leu Gly Phe Cys Leu Gly Val Gly Ser Ala Ile Ala Ser Gly Val Ala
115 120 125
Val Ser Lys Val Leu His Leu Glu Gly Glu Val Asn Lys Ile Lys Ser
130 135 140
Ala Leu Leu Ser Thr Asn Lys Ala Val Val Ser Leu Ser Asn Gly Val
145 150 155 160
Ser Val Leu Val Val Lys Val Leu Asp Leu Lys Asn Tyr Ile Asp Lys
165 170 175
Gln Leu Leu Pro Ile Val Asn Lys Gln Ser Cys Ser Ile Ser Asn Ile
180 185 190
Glu Thr Val Ile Glu Phe Gln Gln Lys Asn Asn Arg Leu Leu Glu Ile
195 200 205
Thr Arg Glu Phe Ser Val Asn Ala Gly Val Thr Thr Pro Val Ser Thr
210 215 220
Tyr Met Leu Thr Asn Ser Glu Leu Leu Ser Leu Ile Asn Asp Met Pro
225 230 235 240
Ile Thr Asn Asp Gln Lys Lys Leu Met Ser Asn Asn Val Gln Ile Val
245 250 255
Arg Gln Gln Ser Tyr Ser Ile Met Ser Ile Ile Lys Glu Glu Val Leu
260 265 270
Ala Tyr Val Val Gln Leu Pro Leu Tyr Gly Val Ile Asp Thr Pro Cys
275 280 285
Trp Lys Leu His Thr Ser Pro Leu Cys Thr Thr Asn Thr Lys Glu Gly
290 295 300
Ser Asn Ile Cys Leu Thr Arg Thr Asp Arg Gly Trp Tyr Cys Asp Asn
305 310 315 320
Ala Gly Ser Val Ser Phe Phe Pro Gln Ala Glu Thr Cys Lys Val Gln
325 330 335
Ser Asn Arg Val Phe Cys Asp Thr Met Asn Ser Cys Thr Leu Pro Ser
340 345 350
Glu Val Asn Leu Cys Asn Thr Asp Ile Phe Asn Pro Lys Tyr Asp Cys
355 360 365
Lys Ile Met Thr Ser Lys Thr Asp Val Ser Ser Ser Val Ile Thr Ser
370 375 380
Leu Gly Ala Ile Val Ser Cys Tyr Gly Lys Thr Lys Cys Thr Ala Ser
385 390 395 400
Asn Lys Asn Arg Gly Ile Ile Lys Thr Phe Ser Asn Gly Cys Asp Tyr
405 410 415
Val Ser Asn Lys Gly Val Asp Thr Val Ser Val Gly Asn Thr Leu Tyr
420 425 430
Tyr Val Asn Lys Gln Glu Gly Lys Ser Leu Tyr Val Lys Gly Glu Pro
435 440 445
Ile Ile Asn Phe Tyr Asp Pro Leu Val Phe Pro Ser Asp Glu Phe Asp
450 455 460
Ala Ser Ile Ser Gln Val Asn Glu Lys Ile Asn Gln Ser Leu Ala Phe
465 470 475 480
Ile Arg Lys Ser Asp Glu Leu Leu
485
<210> 10
<211> 517
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH50-фолдон-метка
<220>
<221> MISC_FEATURE
<222> (1)..(84)
<223> F2
<220>
<221> MISC_FEATURE
<222> (85)..(111)
<223> pep27
<220>
<221> MISC_FEATURE
<222> (112)..(488)
<223> внеклеточный F1 r
<220>
<221> MISC_FEATURE
<222> (489)..(517)
<223> фолдон
<400> 10
Gln Asn Ile Thr Glu Glu Phe Tyr Gln Ser Thr Cys Ser Ala Val Ser
1 5 10 15
Lys Gly Tyr Leu Ser Ala Leu Arg Thr Gly Trp Tyr Thr Ser Val Ile
20 25 30
Thr Ile Glu Leu Ser Asn Ile Lys Glu Asn Lys Cys Asn Gly Thr Asp
35 40 45
Ala Lys Val Lys Leu Ile Lys Gln Glu Leu Asp Lys Tyr Lys Asn Ala
50 55 60
Val Thr Glu Leu Gln Leu Leu Met Gln Ser Thr Pro Ala Thr Asn Asn
65 70 75 80
Arg Ala Arg Arg Glu Leu Pro Arg Phe Met Asn Tyr Thr Leu Asn Asn
85 90 95
Ala Lys Lys Thr Asn Val Thr Leu Ser Lys Lys Arg Lys Asn Asn Phe
100 105 110
Leu Gly Phe Cys Leu Gly Val Gly Ser Ala Ile Ala Ser Gly Val Ala
115 120 125
Val Ser Lys Val Leu His Leu Glu Gly Glu Val Asn Lys Ile Lys Ser
130 135 140
Ala Leu Leu Ser Thr Asn Lys Ala Val Val Ser Leu Ser Asn Gly Val
145 150 155 160
Ser Val Leu Val Val Lys Val Leu Asp Leu Lys Asn Tyr Ile Asp Lys
165 170 175
Gln Leu Leu Pro Ile Val Asn Lys Gln Ser Cys Ser Ile Ser Asn Ile
180 185 190
Glu Thr Val Ile Glu Phe Gln Gln Lys Asn Asn Arg Leu Leu Glu Ile
195 200 205
Thr Arg Glu Phe Ser Val Asn Ala Gly Val Thr Thr Pro Val Ser Thr
210 215 220
Tyr Met Leu Thr Asn Ser Glu Leu Leu Ser Leu Ile Asn Asp Met Pro
225 230 235 240
Ile Thr Asn Asp Gln Lys Lys Leu Met Ser Asn Asn Val Gln Ile Val
245 250 255
Arg Gln Gln Ser Tyr Ser Ile Met Ser Ile Ile Lys Glu Glu Val Leu
260 265 270
Ala Tyr Val Val Gln Leu Pro Leu Tyr Gly Val Ile Asp Thr Pro Cys
275 280 285
Trp Lys Leu His Thr Ser Pro Leu Cys Thr Thr Asn Thr Lys Glu Gly
290 295 300
Ser Asn Ile Cys Leu Thr Arg Thr Asp Arg Gly Trp Tyr Cys Asp Asn
305 310 315 320
Ala Gly Ser Val Ser Phe Phe Pro Gln Ala Glu Thr Cys Lys Val Gln
325 330 335
Ser Asn Arg Val Phe Cys Asp Thr Met Asn Ser Cys Thr Leu Pro Ser
340 345 350
Glu Val Asn Leu Cys Asn Val Asp Ile Phe Asn Pro Lys Tyr Asp Cys
355 360 365
Lys Ile Met Thr Ser Lys Thr Asp Val Ser Ser Ser Val Ile Thr Ser
370 375 380
Leu Gly Ala Ile Val Ser Cys Tyr Gly Lys Thr Lys Cys Thr Ala Ser
385 390 395 400
Asn Lys Asn Arg Gly Ile Ile Lys Thr Phe Ser Asn Gly Cys Asp Tyr
405 410 415
Val Ser Asn Lys Gly Val Asp Thr Val Ser Val Gly Asn Thr Leu Tyr
420 425 430
Tyr Val Asn Lys Gln Glu Gly Lys Ser Leu Tyr Val Lys Gly Glu Pro
435 440 445
Ile Ile Asn Phe Tyr Asp Pro Leu Val Phe Pro Ser Asp Glu Phe Asp
450 455 460
Ala Ser Ile Ser Gln Val Asn Glu Lys Ile Asn Gln Ser Leu Ala Phe
465 470 475 480
Ile Arg Lys Ser Asp Glu Leu Leu Gly Ser Gly Tyr Ile Pro Glu Ala
485 490 495
Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp Val Leu
500 505 510
Leu Ser Thr Phe Leu
515
<210> 11
<211> 517
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH81-фолдон-метка
<220>
<221> MISC_FEATURE
<222> (1)..(84)
<223> F2
<220>
<221> MISC_FEATURE
<222> (85)..(111)
<223> pep27
<220>
<221> MISC_FEATURE
<222> (112)..(488)
<223> внеклеточный F1
<220>
<221> MISC_FEATURE
<222> (489)..(517)
<223> фолдон
<400> 11
Gln Asn Ile Thr Glu Glu Phe Tyr Gln Ser Thr Cys Ser Ala Val Ser
1 5 10 15
Lys Gly Tyr Leu Ser Ala Leu Arg Thr Gly Trp Tyr Thr Ser Val Ile
20 25 30
Thr Ile Met Leu Ser Asn Ile Lys Glu Asn Lys Cys Asn Gly Thr Asp
35 40 45
Ala Lys Val Lys Leu Ile Lys Gln Glu Leu Asp Lys Tyr Lys Asn Ala
50 55 60
Val Thr Glu Leu Gln Leu Leu Met Gln Ser Thr Pro Ala Thr Asn Asn
65 70 75 80
Arg Ala Arg Arg Glu Leu Pro Arg Phe Met Asn Tyr Thr Leu Asn Asn
85 90 95
Ala Lys Lys Thr Asn Val Thr Leu Ser Lys Lys Arg Lys Asn Asn Phe
100 105 110
Leu Gly Phe Cys Leu Gly Val Gly Ser Ala Ile Ala Ser Gly Val Ala
115 120 125
Val Ser Lys Val Leu His Leu Glu Gly Glu Val Asn Lys Ile Lys Ser
130 135 140
Ala Leu Leu Ser Thr Asn Lys Ala Val Val Ser Leu Ser Asn Gly Val
145 150 155 160
Ser Val Leu Val Val Lys Val Leu Asp Leu Lys Asn Tyr Ile Asp Lys
165 170 175
Gln Leu Leu Pro Ile Val Asn Lys Gln Ser Cys Ser Ile Ser Asn Ile
180 185 190
Glu Thr Val Ile Glu Phe Gln Gln Lys Asn Asn Arg Leu Leu Glu Ile
195 200 205
Thr Arg Glu Phe Ser Val Asn Ala Gly Val Thr Thr Pro Val Ser Thr
210 215 220
Tyr Met Leu Thr Asn Ser Glu Leu Leu Ser Leu Ile Asn Asp Met Pro
225 230 235 240
Ile Thr Asn Asp Gln Lys Lys Leu Met Ser Asn Asn Val Gln Ile Val
245 250 255
Arg Gln Gln Ser Tyr Ser Ile Met Ser Ile Ile Lys Glu Glu Val Leu
260 265 270
Ala Tyr Val Val Gln Leu Pro Leu Tyr Gly Val Ile Asp Thr Pro Cys
275 280 285
Trp Lys Leu His Thr Ser Pro Leu Cys Thr Thr Asn Thr Lys Glu Gly
290 295 300
Ser Asn Ile Cys Leu Thr Arg Thr Asp Arg Gly Trp Tyr Cys Asp Asn
305 310 315 320
Ala Gly Ser Val Ser Phe Phe Pro Gln Ala Glu Thr Cys Lys Val Gln
325 330 335
Ser Asn Arg Val Phe Cys Asp Thr Met Asn Ser Cys Thr Leu Pro Ser
340 345 350
Glu Val Asn Leu Cys Asn Val Asp Ile Phe Asn Pro Lys Tyr Asp Cys
355 360 365
Lys Ile Met Thr Ser Lys Thr Asp Val Ser Ser Ser Val Ile Thr Ser
370 375 380
Leu Gly Ala Ile Val Ser Cys Tyr Gly Lys Thr Lys Cys Thr Ala Ser
385 390 395 400
Asn Lys Asn Arg Gly Ile Ile Lys Thr Phe Ser Asn Gly Cys Asp Tyr
405 410 415
Val Ser Asn Lys Gly Val Asp Thr Val Ser Val Gly Asn Thr Leu Tyr
420 425 430
Tyr Val Asn Lys Gln Glu Gly Lys Ser Leu Tyr Val Lys Gly Glu Pro
435 440 445
Ile Ile Asn Phe Tyr Asp Pro Leu Val Phe Pro Ser Asp Glu Phe Asp
450 455 460
Ala Ser Ile Ser Gln Val Asn Glu Lys Ile Asn Gln Ser Leu Ala Phe
465 470 475 480
Ile Arg Lys Ser Asp Glu Leu Leu Gly Ser Gly Tyr Ile Pro Glu Ala
485 490 495
Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp Val Leu
500 505 510
Leu Ser Thr Phe Leu
515
<210> 12
<211> 517
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH82-фолдон-метка
<220>
<221> MISC_FEATURE
<222> (1)..(84)
<223> F2
<220>
<221> MISC_FEATURE
<222> (85)..(111)
<223> pep27
<220>
<221> MISC_FEATURE
<222> (112)..(488)
<223> внеклеточный F1
<220>
<221> MISC_FEATURE
<222> (489)..(517)
<223> фолдон
<400> 12
Gln Asn Ile Thr Glu Glu Phe Tyr Gln Ser Thr Cys Ser Ala Val Ser
1 5 10 15
Arg Gly Tyr Leu Ser Ala Leu Arg Thr Gly Trp Tyr Thr Ser Val Ile
20 25 30
Thr Ile Glu Leu Ser Asn Ile Lys Glu Asn Lys Cys Asn Gly Thr Asp
35 40 45
Ala Lys Val Lys Leu Ile Lys Gln Glu Leu Asp Lys Tyr Lys Asn Ala
50 55 60
Val Thr Glu Leu Gln Leu Leu Met Gln Ser Thr Pro Ala Thr Asn Asn
65 70 75 80
Arg Ala Arg Arg Glu Leu Pro Arg Phe Met Asn Tyr Thr Leu Asn Asn
85 90 95
Ala Lys Lys Thr Asn Val Thr Leu Ser Lys Lys Arg Lys Asn Asn Phe
100 105 110
Leu Gly Phe Cys Leu Gly Val Gly Ser Ala Ile Ala Ser Gly Val Ala
115 120 125
Val Ser Lys Val Leu His Leu Glu Gly Glu Val Asn Lys Ile Lys Ser
130 135 140
Ala Leu Leu Ser Thr Asn Lys Ala Val Val Ser Leu Ser Asn Gly Val
145 150 155 160
Ser Val Leu Val Val Lys Val Leu Asp Leu Lys Asn Tyr Ile Asp Lys
165 170 175
Gln Leu Leu Pro Ile Val Asn Lys Gln Ser Cys Ser Ile Ser Asn Ile
180 185 190
Glu Thr Val Ile Glu Phe Gln Gln Lys Asn Asn Arg Leu Leu Glu Ile
195 200 205
Thr Arg Glu Phe Ser Val Asn Ala Gly Val Thr Thr Pro Val Ser Thr
210 215 220
Tyr Met Leu Thr Asn Ser Glu Leu Leu Ser Leu Ile Asn Asp Met Pro
225 230 235 240
Ile Thr Asn Asp Gln Lys Lys Leu Met Ser Asn Asn Val Gln Ile Val
245 250 255
Arg Gln Gln Ser Tyr Ser Ile Met Ser Ile Ile Lys Glu Glu Val Leu
260 265 270
Ala Tyr Val Val Gln Leu Pro Leu Tyr Gly Val Ile Asp Thr Pro Cys
275 280 285
Trp Lys Leu His Thr Ser Pro Leu Cys Thr Thr Asn Thr Lys Glu Gly
290 295 300
Ser Asn Ile Cys Leu Thr Arg Thr Asp Arg Gly Trp Tyr Cys Asp Asn
305 310 315 320
Ala Gly Ser Val Ser Phe Phe Pro Gln Ala Glu Thr Cys Lys Val Gln
325 330 335
Ser Asn Arg Val Phe Cys Asp Thr Met Asn Ser Cys Thr Leu Pro Ser
340 345 350
Glu Val Asn Leu Cys Asn Thr Asp Ile Phe Asn Pro Lys Tyr Asp Cys
355 360 365
Lys Ile Met Thr Ser Lys Thr Asp Val Ser Ser Ser Val Ile Thr Ser
370 375 380
Leu Gly Ala Ile Val Ser Cys Tyr Gly Lys Thr Lys Cys Thr Ala Ser
385 390 395 400
Asn Lys Asn Arg Gly Ile Ile Lys Thr Phe Ser Asn Gly Cys Asp Tyr
405 410 415
Val Ser Asn Lys Gly Val Asp Thr Val Ser Val Gly Asn Thr Leu Tyr
420 425 430
Tyr Val Asn Lys Gln Glu Gly Lys Ser Leu Tyr Val Lys Gly Glu Pro
435 440 445
Ile Ile Asn Phe Tyr Asp Pro Leu Val Phe Pro Ser Asp Glu Phe Asp
450 455 460
Ala Ser Ile Ser Gln Val Asn Glu Lys Ile Asn Gln Ser Leu Ala Phe
465 470 475 480
Ile Arg Lys Ser Asp Glu Leu Leu Gly Ser Gly Tyr Ile Pro Glu Ala
485 490 495
Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp Val Leu
500 505 510
Leu Ser Thr Phe Leu
515
<210> 13
<211> 517
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH85-фолдон-метка
<220>
<221> MISC_FEATURE
<222> (1)..(84)
<223> F2
<220>
<221> MISC_FEATURE
<222> (85)..(111)
<223> pep27
<220>
<221> MISC_FEATURE
<222> (112)..(488)
<223> внеклеточный F1
<220>
<221> MISC_FEATURE
<222> (489)..(517)
<223> фолдон
<400> 13
Gln Asn Ile Thr Glu Glu Phe Tyr Gln Ser Thr Cys Ser Ala Val Ser
1 5 10 15
Arg Gly Tyr Leu Ser Ala Leu Arg Thr Gly Trp Tyr Thr Ser Val Ile
20 25 30
Thr Ile Met Leu Ser Asn Ile Lys Glu Asn Lys Cys Asn Gly Thr Asp
35 40 45
Ala Lys Val Lys Leu Ile Lys Gln Glu Leu Asp Lys Tyr Lys Asn Ala
50 55 60
Val Thr Glu Leu Gln Leu Leu Met Gln Ser Thr Pro Ala Thr Asn Asn
65 70 75 80
Arg Ala Arg Arg Glu Leu Pro Arg Phe Met Asn Tyr Thr Leu Asn Asn
85 90 95
Ala Lys Lys Thr Asn Val Thr Leu Ser Lys Lys Arg Lys Asn Asn Phe
100 105 110
Leu Gly Phe Cys Leu Gly Val Gly Ser Ala Ile Ala Ser Gly Val Ala
115 120 125
Val Ser Lys Val Leu His Leu Glu Gly Glu Val Asn Lys Ile Lys Ser
130 135 140
Ala Leu Leu Ser Thr Asn Lys Ala Val Val Ser Leu Ser Asn Gly Val
145 150 155 160
Ser Val Leu Val Val Lys Val Leu Asp Leu Lys Asn Tyr Ile Asp Lys
165 170 175
Gln Leu Leu Pro Ile Val Asn Lys Gln Ser Cys Ser Ile Ser Asn Ile
180 185 190
Glu Thr Val Ile Glu Phe Gln Gln Lys Asn Asn Arg Leu Leu Glu Ile
195 200 205
Thr Arg Glu Phe Ser Val Asn Ala Gly Val Thr Thr Pro Val Ser Thr
210 215 220
Tyr Met Leu Thr Asn Ser Glu Leu Leu Ser Leu Ile Asn Asp Met Pro
225 230 235 240
Ile Thr Asn Asp Gln Lys Lys Leu Met Ser Asn Asn Val Gln Ile Val
245 250 255
Arg Gln Gln Ser Tyr Ser Ile Met Ser Ile Ile Lys Glu Glu Val Leu
260 265 270
Ala Tyr Val Val Gln Leu Pro Leu Tyr Gly Val Ile Asp Thr Pro Cys
275 280 285
Trp Lys Leu His Thr Ser Pro Leu Cys Thr Thr Asn Thr Lys Glu Gly
290 295 300
Ser Asn Ile Cys Leu Thr Arg Thr Asp Arg Gly Trp Tyr Cys Asp Asn
305 310 315 320
Ala Gly Ser Val Ser Phe Phe Pro Gln Ala Glu Thr Cys Lys Val Gln
325 330 335
Ser Asn Arg Val Phe Cys Asp Thr Met Asn Ser Cys Thr Leu Pro Ser
340 345 350
Glu Val Asn Leu Cys Asn Thr Asp Ile Phe Asn Pro Lys Tyr Asp Cys
355 360 365
Lys Ile Met Thr Ser Lys Thr Asp Val Ser Ser Ser Val Ile Thr Ser
370 375 380
Leu Gly Ala Ile Val Ser Cys Tyr Gly Lys Thr Lys Cys Thr Ala Ser
385 390 395 400
Asn Lys Asn Arg Gly Ile Ile Lys Thr Phe Ser Asn Gly Cys Asp Tyr
405 410 415
Val Ser Asn Lys Gly Val Asp Thr Val Ser Val Gly Asn Thr Leu Tyr
420 425 430
Tyr Val Asn Lys Gln Glu Gly Lys Ser Leu Tyr Val Lys Gly Glu Pro
435 440 445
Ile Ile Asn Phe Tyr Asp Pro Leu Val Phe Pro Ser Asp Glu Phe Asp
450 455 460
Ala Ser Ile Ser Gln Val Asn Glu Lys Ile Asn Gln Ser Leu Ala Phe
465 470 475 480
Ile Arg Lys Ser Asp Glu Leu Leu Gly Ser Gly Tyr Ile Pro Glu Ala
485 490 495
Pro Arg Asp Gly Gln Ala Tyr Val Arg Lys Asp Gly Glu Trp Val Leu
500 505 510
Leu Ser Thr Phe Leu
515
<210> 14
<211> 1626
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Последовательность, кодирующая FH50-фолдон-метку
<400> 14
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agcaaagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcgag 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaatg tagacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gatcaggcta cattcccgaa 1560
gctccaaggg acggacaagc atacgtacga aaagatggcg aatgggtact cctctcaacg 1620
tttctg 1626
<210> 15
<211> 1626
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Последовательность, кодирующая FH81-фолдон-метку
<400> 15
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agcaaagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcatg 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaatg tagacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gatcaggcta cattcccgaa 1560
gctccaaggg acggacaagc atacgtacga aaagatggcg aatgggtact cctctcaacg 1620
tttctg 1626
<210> 16
<211> 1626
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Последовательность, кодирующая FH82-фолдон-метку
<400> 16
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agccgagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcgag 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaata ccgacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gatcaggcta cattcccgaa 1560
gctccaaggg acggacaagc atacgtacga aaagatggcg aatgggtact cctctcaacg 1620
tttctg 1626
<210> 17
<211> 1626
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Последовательность, кодирующая FH85-фолдон-метку
<400> 17
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agccgagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcatg 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaata ccgacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gatcaggcta cattcccgaa 1560
gctccaaggg acggacaagc atacgtacga aaagatggcg aatgggtact cctctcaacg 1620
tttctg 1626
<210> 18
<211> 6
<212> PRT
<213> Искусственная последовательность
<220>
<223> линкер GS
<400> 18
Gly Ser Gly Ser Gly Ser
1 5
<210> 19
<211> 10
<212> PRT
<213> Искусственная последовательность
<220>
<223> линкер GS
<400> 19
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10
<210> 20
<211> 15
<212> PRT
<213> Искусственная последовательность
<220>
<223> линкер GS
<400> 20
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 21
<211> 29
<212> PRT
<213> Искусственная последовательность
<220>
<223> фолдон
<400> 21
Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val
1 5 10 15
Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu
20 25
<210> 22
<211> 20
<212> PRT
<213> Искусственная последовательность
<220>
<223> метка
<400> 22
Leu Val Pro Arg Gly Ser His His His His His His Trp Ser His Pro
1 5 10 15
Gln Phe Glu Lys
20
<210> 23
<211> 562
<212> PRT
<213> Искусственная последовательность
<220>
<223> DS-Cav1
<400> 23
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Cys Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Phe Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Leu Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Cys Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Leu Val
530 535 540
Pro Arg Gly Ser His His His His His His Trp Ser His Pro Gln Phe
545 550 555 560
Glu Lys
<210> 24
<211> 521
<212> PRT
<213> Искусственная последовательность
<220>
<223> delta FP furinwt Fecto
<400> 24
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Ser Ala Ile Ala Ser Gly Val Ala
130 135 140
Val Ser Lys Val Leu His Leu Glu Gly Glu Val Asn Lys Ile Lys Ser
145 150 155 160
Ala Leu Leu Ser Thr Asn Lys Ala Val Val Ser Leu Ser Asn Gly Val
165 170 175
Ser Val Leu Thr Ser Lys Val Leu Asp Leu Lys Asn Tyr Ile Asp Lys
180 185 190
Gln Leu Leu Pro Ile Val Asn Lys Gln Ser Cys Ser Ile Ser Asn Ile
195 200 205
Glu Thr Val Ile Glu Phe Gln Gln Lys Asn Asn Arg Leu Leu Glu Ile
210 215 220
Thr Arg Glu Phe Ser Val Asn Ala Gly Val Thr Thr Pro Val Ser Thr
225 230 235 240
Tyr Met Leu Thr Asn Ser Glu Leu Leu Ser Leu Ile Asn Asp Met Pro
245 250 255
Ile Thr Asn Asp Gln Lys Lys Leu Met Ser Asn Asn Val Gln Ile Val
260 265 270
Arg Gln Gln Ser Tyr Ser Ile Met Ser Ile Ile Lys Glu Glu Val Leu
275 280 285
Ala Tyr Val Val Gln Leu Pro Leu Tyr Gly Val Ile Asp Thr Pro Cys
290 295 300
Trp Lys Leu His Thr Ser Pro Leu Cys Thr Thr Asn Thr Lys Glu Gly
305 310 315 320
Ser Asn Ile Cys Leu Thr Arg Thr Asp Arg Gly Trp Tyr Cys Asp Asn
325 330 335
Ala Gly Ser Val Ser Phe Phe Pro Gln Ala Glu Thr Cys Lys Val Gln
340 345 350
Ser Asn Arg Val Phe Cys Asp Thr Met Asn Ser Leu Thr Leu Pro Ser
355 360 365
Glu Val Asn Leu Cys Asn Val Asp Ile Phe Asn Pro Lys Tyr Asp Cys
370 375 380
Lys Ile Met Thr Ser Lys Thr Asp Val Ser Ser Ser Val Ile Thr Ser
385 390 395 400
Leu Gly Ala Ile Val Ser Cys Tyr Gly Lys Thr Lys Cys Thr Ala Ser
405 410 415
Asn Lys Asn Arg Gly Ile Ile Lys Thr Phe Ser Asn Gly Cys Asp Tyr
420 425 430
Val Ser Asn Lys Gly Val Asp Thr Val Ser Val Gly Asn Thr Leu Tyr
435 440 445
Tyr Val Asn Lys Gln Glu Gly Lys Ser Leu Tyr Val Lys Gly Glu Pro
450 455 460
Ile Ile Asn Phe Tyr Asp Pro Leu Val Phe Pro Ser Asp Glu Phe Asp
465 470 475 480
Ala Ser Ile Ser Gln Val Asn Glu Lys Ile Asn Gln Ser Leu Ala Phe
485 490 495
Ile Arg Lys Ser Asp Glu Leu Leu His Asn Val Asn Ala Gly Lys Ser
500 505 510
Thr Thr Asn His His His His His His
515 520
<210> 25
<211> 6
<212> PRT
<213> Искусственная последовательность
<220>
<223> His-метка
<400> 25
His His His His His His
1 5
<210> 26
<211> 8
<212> PRT
<213> Искусственная последовательность
<220>
<223> Strep-метка II
<400> 26
Trp Ser His Pro Gln Phe Glu Lys
1 5
<210> 27
<211> 5
<212> PRT
<213> Искусственная последовательность
<220>
<223> линкер GS
<400> 27
Gly Gly Gly Gly Ser
1 5
<210> 28
<211> 8
<212> PRT
<213> Искусственная последовательность
<220>
<223> FLAG-метка
<400> 28
Asp Tyr Lys Asp Asp Asp Asp Lys
1 5
<210> 29
<211> 11
<212> PRT
<213> Искусственная последовательность
<220>
<223> GGS+Flag
<400> 29
Gly Gly Ser Asp Tyr Lys Asp Asp Asp Asp Lys
1 5 10
<210> 30
<211> 227
<212> PRT
<213> Искусственная последовательность
<220>
<223> Fc
<400> 30
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
1 5 10 15
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
20 25 30
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
35 40 45
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
50 55 60
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
65 70 75 80
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
85 90 95
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
100 105 110
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
115 120 125
Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser
130 135 140
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
145 150 155 160
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
165 170 175
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
180 185 190
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
195 200 205
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
210 215 220
Pro Gly Lys
225
<210> 31
<211> 769
<212> PRT
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-Fc
<400> 31
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Cys Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Phe Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Leu Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Cys Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Asp Lys
530 535 540
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
545 550 555 560
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
565 570 575
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
580 585 590
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
595 600 605
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
610 615 620
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
625 630 635 640
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
645 650 655
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
660 665 670
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
675 680 685
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
690 695 700
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
705 710 715 720
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
725 730 735
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
740 745 750
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
755 760 765
Lys
<210> 32
<211> 769
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_82-Fc
<400> 32
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Arg Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Asn Asn Phe Leu Gly Phe Cys Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Ser Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Val Val Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Val Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Ser Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Cys Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Thr
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Asp Lys
530 535 540
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
545 550 555 560
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
565 570 575
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
580 585 590
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
595 600 605
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
610 615 620
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
625 630 635 640
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
645 650 655
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
660 665 670
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
675 680 685
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
690 695 700
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
705 710 715 720
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
725 730 735
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
740 745 750
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
755 760 765
Lys
<210> 33
<211> 769
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_85-Fc
<400> 33
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Arg Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Met Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Asn Asn Phe Leu Gly Phe Cys Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Ser Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Val Val Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Val Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Ser Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Cys Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Thr
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Asp Lys
530 535 540
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
545 550 555 560
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
565 570 575
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
580 585 590
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
595 600 605
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
610 615 620
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
625 630 635 640
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
645 650 655
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
660 665 670
Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr
675 680 685
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
690 695 700
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
705 710 715 720
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
725 730 735
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
740 745 750
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
755 760 765
Lys
<210> 34
<211> 789
<212> PRT
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-4GS-Fc
<400> 34
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Cys Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Phe Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Leu Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Cys Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Gly
530 535 540
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
545 550 555 560
Gly Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
565 570 575
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
580 585 590
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
595 600 605
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
610 615 620
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
625 630 635 640
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
645 650 655
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
660 665 670
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
675 680 685
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
690 695 700
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
705 710 715 720
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
725 730 735
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
740 745 750
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
755 760 765
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
770 775 780
Leu Ser Pro Gly Lys
785
<210> 35
<211> 779
<212> PRT
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-2GS-Fc
<400> 35
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Cys Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Phe Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Leu Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Cys Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Gly
530 535 540
Gly Gly Ser Gly Gly Gly Gly Ser Asp Lys Thr His Thr Cys Pro Pro
545 550 555 560
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
565 570 575
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
580 585 590
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
595 600 605
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
610 615 620
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
625 630 635 640
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
645 650 655
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
660 665 670
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
675 680 685
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
690 695 700
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
705 710 715 720
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
725 730 735
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
740 745 750
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
755 760 765
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
770 775
<210> 36
<211> 774
<212> PRT
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-1GS-Fc
<400> 36
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Cys Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Phe Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Leu Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Cys Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Gly
530 535 540
Gly Gly Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
545 550 555 560
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
565 570 575
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
580 585 590
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
595 600 605
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
610 615 620
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
625 630 635 640
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
645 650 655
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
660 665 670
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
675 680 685
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
690 695 700
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
705 710 715 720
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
725 730 735
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
740 745 750
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
755 760 765
Ser Leu Ser Pro Gly Lys
770
<210> 37
<211> 789
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_85-4GS-Fc
<400> 37
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Arg Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Met Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Asn Asn Phe Leu Gly Phe Cys Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Ser Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Val Val Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Val Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Ser Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Cys Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Thr
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Gly
530 535 540
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
545 550 555 560
Gly Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
565 570 575
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
580 585 590
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
595 600 605
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
610 615 620
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
625 630 635 640
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
645 650 655
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
660 665 670
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
675 680 685
Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln
690 695 700
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
705 710 715 720
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
725 730 735
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
740 745 750
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
755 760 765
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
770 775 780
Leu Ser Pro Gly Lys
785
<210> 38
<211> 779
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_85-2GS-Fc
<400> 38
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Arg Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Met Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Asn Asn Phe Leu Gly Phe Cys Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Ser Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Val Val Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Val Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Ser Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Cys Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Thr
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Gly
530 535 540
Gly Gly Ser Gly Gly Gly Gly Ser Asp Lys Thr His Thr Cys Pro Pro
545 550 555 560
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
565 570 575
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
580 585 590
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
595 600 605
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
610 615 620
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
625 630 635 640
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
645 650 655
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
660 665 670
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
675 680 685
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
690 695 700
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
705 710 715 720
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
725 730 735
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
740 745 750
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
755 760 765
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
770 775
<210> 39
<211> 774
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_85-1GS-Fc
<400> 39
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Arg Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Met Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Asn Asn Phe Leu Gly Phe Cys Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Ser Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Val Val Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Val Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Ser Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Cys Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Thr
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Gly
530 535 540
Gly Gly Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
545 550 555 560
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
565 570 575
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
580 585 590
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
595 600 605
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
610 615 620
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
625 630 635 640
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
645 650 655
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
660 665 670
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
675 680 685
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
690 695 700
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
705 710 715 720
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
725 730 735
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
740 745 750
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
755 760 765
Ser Leu Ser Pro Gly Lys
770
<210> 40
<211> 21
<212> PRT
<213> Искусственная последовательность
<220>
<223> CC02
<400> 40
Ile Ala Leu Ala Leu Glu Lys Ile Ala Leu Ala Leu Glu Lys Ile Ala
1 5 10 15
Leu Ala Leu Glu Lys
20
<210> 41
<211> 21
<212> PRT
<213> Искусственная последовательность
<220>
<223> CC03
<400> 41
Ile Lys Leu Glu Leu Glu Lys Ile Lys Leu Glu Leu Glu Lys Ile Lys
1 5 10 15
Leu Glu Leu Glu Lys
20
<210> 42
<211> 21
<212> PRT
<213> Искусственная последовательность
<220>
<223> CC07
<400> 42
Leu His Leu His Ile Glu Lys Leu His Leu His Ile Glu Lys Leu His
1 5 10 15
Leu His Ile Glu Lys
20
<210> 43
<211> 21
<212> PRT
<213> Искусственная последовательность
<220>
<223> CCO08
<400> 43
Leu Lys Leu Glu Ile His His Leu Lys Leu Glu Ile His His Leu Lys
1 5 10 15
Leu Glu Ile His His
20
<210> 44
<211> 571
<212> PRT
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-CC02
<400> 44
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Cys Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Phe Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Leu Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Cys Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Ile Ala
530 535 540
Leu Ala Leu Glu Lys Ile Ala Leu Ala Leu Glu Lys Ile Ala Leu Ala
545 550 555 560
Leu Glu Lys Asp Tyr Lys Asp Asp Asp Asp Lys
565 570
<210> 45
<211> 571
<212> PRT
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-CC03
<400> 45
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Cys Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Phe Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Leu Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Cys Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Ile Lys
530 535 540
Leu Glu Leu Glu Lys Ile Lys Leu Glu Leu Glu Lys Ile Lys Leu Glu
545 550 555 560
Leu Glu Lys Asp Tyr Lys Asp Asp Asp Asp Lys
565 570
<210> 46
<211> 571
<212> PRT
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-CC07
<400> 46
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Cys Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Phe Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Leu Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Cys Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Leu His
530 535 540
Leu His Ile Glu Lys Leu His Leu His Ile Glu Lys Leu His Leu His
545 550 555 560
Ile Glu Lys Asp Tyr Lys Asp Asp Asp Asp Lys
565 570
<210> 47
<211> 571
<212> PRT
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-CC08
<400> 47
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Cys Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Phe Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Leu Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Cys Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Leu Lys
530 535 540
Leu Glu Ile His His Leu Lys Leu Glu Ile His His Leu Lys Leu Glu
545 550 555 560
Ile His His Asp Tyr Lys Asp Asp Asp Asp Lys
565 570
<210> 48
<211> 571
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_82-CC07
<400> 48
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Arg Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Asn Asn Phe Leu Gly Phe Cys Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Ser Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Val Val Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Val Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Ser Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Cys Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Thr
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Leu His
530 535 540
Leu His Ile Glu Lys Leu His Leu His Ile Glu Lys Leu His Leu His
545 550 555 560
Ile Glu Lys Asp Tyr Lys Asp Asp Asp Asp Lys
565 570
<210> 49
<211> 571
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_85-CC07
<400> 49
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Arg Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Met Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Asn Asn Phe Leu Gly Phe Cys Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Ser Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Val Val Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Val Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Ser Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Cys Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Thr
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Leu His
530 535 540
Leu His Ile Glu Lys Leu His Leu His Ile Glu Lys Leu His Leu His
545 550 555 560
Ile Glu Lys Asp Tyr Lys Asp Asp Asp Asp Lys
565 570
<210> 50
<211> 149
<212> PRT
<213> Искусственная последовательность
<220>
<223> Самоассоциирующийся домен HBcAg
<400> 50
Met Asp Ile Asp Pro Tyr Lys Glu Phe Gly Ala Ser Val Glu Leu Leu
1 5 10 15
Ser Phe Leu Pro Ser Asp Phe Phe Pro Ser Ile Arg Asp Leu Leu Asp
20 25 30
Thr Ala Ser Ala Leu Tyr Arg Glu Ala Leu Glu Ser Pro Glu His Cys
35 40 45
Ser Pro His His Thr Ala Leu Arg Gln Ala Ile Leu Cys Trp Gly Glu
50 55 60
Leu Met Asn Leu Ala Thr Trp Val Gly Ser Asn Leu Glu Asp Pro Ala
65 70 75 80
Ser Arg Glu Leu Val Val Ser Tyr Val Asn Val Asn Met Gly Leu Lys
85 90 95
Ile Arg Gln Leu Leu Trp Phe His Ile Ser Cys Leu Thr Phe Gly Arg
100 105 110
Glu Thr Val Leu Glu Tyr Leu Val Ser Phe Gly Val Trp Ile Arg Thr
115 120 125
Pro Pro Ala Ala Arg Pro Pro Asn Ala Pro Ile Leu Ser Thr Leu Pro
130 135 140
Glu Thr Thr Val Val
145
<210> 51
<211> 175
<212> PRT
<213> Искусственная последовательность
<220>
<223> GS+самоассоциирующийся домен HBcAg+His
<400> 51
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser Met Asp Ile Asp Pro Tyr Lys Glu Phe Gly Ala Ser
20 25 30
Val Glu Leu Leu Ser Phe Leu Pro Ser Asp Phe Phe Pro Ser Ile Arg
35 40 45
Asp Leu Leu Asp Thr Ala Ser Ala Leu Tyr Arg Glu Ala Leu Glu Ser
50 55 60
Pro Glu His Cys Ser Pro His His Thr Ala Leu Arg Gln Ala Ile Leu
65 70 75 80
Cys Trp Gly Glu Leu Met Asn Leu Ala Thr Trp Val Gly Ser Asn Leu
85 90 95
Glu Asp Pro Ala Ser Arg Glu Leu Val Val Ser Tyr Val Asn Val Asn
100 105 110
Met Gly Leu Lys Ile Arg Gln Leu Leu Trp Phe His Ile Ser Cys Leu
115 120 125
Thr Phe Gly Arg Glu Thr Val Leu Glu Tyr Leu Val Ser Phe Gly Val
130 135 140
Trp Ile Arg Thr Pro Pro Ala Ala Arg Pro Pro Asn Ala Pro Ile Leu
145 150 155 160
Ser Thr Leu Pro Glu Thr Thr Val Val His His His His His His
165 170 175
<210> 52
<211> 70
<212> PRT
<213> Искусственная последовательность
<220>
<223> C-концевая сторона самоассоциирующегося домена HBcAg
<400> 52
Ala Ser Arg Glu Leu Val Val Ser Tyr Val Asn Val Asn Met Gly Leu
1 5 10 15
Lys Ile Arg Gln Leu Leu Trp Phe His Ile Ser Ala Leu Thr Phe Gly
20 25 30
Arg Glu Thr Val Leu Glu Tyr Leu Val Ser Phe Gly Val Trp Ile Arg
35 40 45
Thr Pro Pro Ala Ala Arg Pro Pro Asn Ala Pro Ile Leu Ser Thr Leu
50 55 60
Pro Glu Thr Thr Val Val
65 70
<210> 53
<211> 65
<212> PRT
<213> Искусственная последовательность
<220>
<223> N-концевая сторона самоассоциирующегося домена HBcAg
<400> 53
Gly Ala Ser Val Glu Leu Leu Ser Phe Leu Pro Ser Asp Phe Phe Pro
1 5 10 15
Ser Ile Arg Asp Leu Leu Asp Thr Ala Ser Ala Leu Tyr Arg Glu Ala
20 25 30
Leu Glu Ser Pro Glu His Ser Ser Pro His His Thr Ala Leu Arg Gln
35 40 45
Ala Ile Leu Cys Trp Gly Glu Leu Met Asn Leu Ala Thr Trp Val Gly
50 55 60
Ser
65
<210> 54
<211> 186
<212> PRT
<213> Искусственная последовательность
<220>
<223> HBcCN
<400> 54
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser Ala Ser Arg Glu Leu Val Val Ser Tyr Val Asn Val
20 25 30
Asn Met Gly Leu Lys Ile Arg Gln Leu Leu Trp Phe His Ile Ser Ala
35 40 45
Leu Thr Phe Gly Arg Glu Thr Val Leu Glu Tyr Leu Val Ser Phe Gly
50 55 60
Val Trp Ile Arg Thr Pro Pro Ala Ala Arg Pro Pro Asn Ala Pro Ile
65 70 75 80
Leu Ser Thr Leu Pro Glu Thr Thr Val Val Gly Gly Gly Gly Ser Gly
85 90 95
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Ala
100 105 110
Ser Val Glu Leu Leu Ser Phe Leu Pro Ser Asp Phe Phe Pro Ser Ile
115 120 125
Arg Asp Leu Leu Asp Thr Ala Ser Ala Leu Tyr Arg Glu Ala Leu Glu
130 135 140
Ser Pro Glu His Ser Ser Pro His His Thr Ala Leu Arg Gln Ala Ile
145 150 155 160
Leu Cys Trp Gly Glu Leu Met Asn Leu Ala Thr Trp Val Gly Ser Gly
165 170 175
Gly Ser Asp Tyr Lys Asp Asp Asp Asp Lys
180 185
<210> 55
<211> 728
<212> PRT
<213> Искусственная последовательность
<220>
<223> F_DC-Cav1-HBcCN
<400> 55
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Lys Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Glu Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Arg Arg Phe Leu Gly Phe Leu Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Cys Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Thr Phe Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Leu Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Cys Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Leu Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Val
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Gly
530 535 540
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
545 550 555 560
Gly Ser Ala Ser Arg Glu Leu Val Val Ser Tyr Val Asn Val Asn Met
565 570 575
Gly Leu Lys Ile Arg Gln Leu Leu Trp Phe His Ile Ser Ala Leu Thr
580 585 590
Phe Gly Arg Glu Thr Val Leu Glu Tyr Leu Val Ser Phe Gly Val Trp
595 600 605
Ile Arg Thr Pro Pro Ala Ala Arg Pro Pro Asn Ala Pro Ile Leu Ser
610 615 620
Thr Leu Pro Glu Thr Thr Val Val Gly Gly Gly Gly Ser Gly Gly Gly
625 630 635 640
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Ala Ser Val
645 650 655
Glu Leu Leu Ser Phe Leu Pro Ser Asp Phe Phe Pro Ser Ile Arg Asp
660 665 670
Leu Leu Asp Thr Ala Ser Ala Leu Tyr Arg Glu Ala Leu Glu Ser Pro
675 680 685
Glu His Ser Ser Pro His His Thr Ala Leu Arg Gln Ala Ile Leu Cys
690 695 700
Trp Gly Glu Leu Met Asn Leu Ala Thr Trp Val Gly Ser Gly Gly Ser
705 710 715 720
Asp Tyr Lys Asp Asp Asp Asp Lys
725
<210> 56
<211> 728
<212> PRT
<213> Искусственная последовательность
<220>
<223> FH_85-HBcCN
<400> 56
Met Glu Leu Leu Ile Leu Lys Ala Asn Ala Ile Thr Thr Ile Leu Thr
1 5 10 15
Ala Val Thr Phe Cys Phe Ala Ser Gly Gln Asn Ile Thr Glu Glu Phe
20 25 30
Tyr Gln Ser Thr Cys Ser Ala Val Ser Arg Gly Tyr Leu Ser Ala Leu
35 40 45
Arg Thr Gly Trp Tyr Thr Ser Val Ile Thr Ile Met Leu Ser Asn Ile
50 55 60
Lys Glu Asn Lys Cys Asn Gly Thr Asp Ala Lys Val Lys Leu Ile Lys
65 70 75 80
Gln Glu Leu Asp Lys Tyr Lys Asn Ala Val Thr Glu Leu Gln Leu Leu
85 90 95
Met Gln Ser Thr Pro Ala Thr Asn Asn Arg Ala Arg Arg Glu Leu Pro
100 105 110
Arg Phe Met Asn Tyr Thr Leu Asn Asn Ala Lys Lys Thr Asn Val Thr
115 120 125
Leu Ser Lys Lys Arg Lys Asn Asn Phe Leu Gly Phe Cys Leu Gly Val
130 135 140
Gly Ser Ala Ile Ala Ser Gly Val Ala Val Ser Lys Val Leu His Leu
145 150 155 160
Glu Gly Glu Val Asn Lys Ile Lys Ser Ala Leu Leu Ser Thr Asn Lys
165 170 175
Ala Val Val Ser Leu Ser Asn Gly Val Ser Val Leu Val Val Lys Val
180 185 190
Leu Asp Leu Lys Asn Tyr Ile Asp Lys Gln Leu Leu Pro Ile Val Asn
195 200 205
Lys Gln Ser Cys Ser Ile Ser Asn Ile Glu Thr Val Ile Glu Phe Gln
210 215 220
Gln Lys Asn Asn Arg Leu Leu Glu Ile Thr Arg Glu Phe Ser Val Asn
225 230 235 240
Ala Gly Val Thr Thr Pro Val Ser Thr Tyr Met Leu Thr Asn Ser Glu
245 250 255
Leu Leu Ser Leu Ile Asn Asp Met Pro Ile Thr Asn Asp Gln Lys Lys
260 265 270
Leu Met Ser Asn Asn Val Gln Ile Val Arg Gln Gln Ser Tyr Ser Ile
275 280 285
Met Ser Ile Ile Lys Glu Glu Val Leu Ala Tyr Val Val Gln Leu Pro
290 295 300
Leu Tyr Gly Val Ile Asp Thr Pro Cys Trp Lys Leu His Thr Ser Pro
305 310 315 320
Leu Cys Thr Thr Asn Thr Lys Glu Gly Ser Asn Ile Cys Leu Thr Arg
325 330 335
Thr Asp Arg Gly Trp Tyr Cys Asp Asn Ala Gly Ser Val Ser Phe Phe
340 345 350
Pro Gln Ala Glu Thr Cys Lys Val Gln Ser Asn Arg Val Phe Cys Asp
355 360 365
Thr Met Asn Ser Cys Thr Leu Pro Ser Glu Val Asn Leu Cys Asn Thr
370 375 380
Asp Ile Phe Asn Pro Lys Tyr Asp Cys Lys Ile Met Thr Ser Lys Thr
385 390 395 400
Asp Val Ser Ser Ser Val Ile Thr Ser Leu Gly Ala Ile Val Ser Cys
405 410 415
Tyr Gly Lys Thr Lys Cys Thr Ala Ser Asn Lys Asn Arg Gly Ile Ile
420 425 430
Lys Thr Phe Ser Asn Gly Cys Asp Tyr Val Ser Asn Lys Gly Val Asp
435 440 445
Thr Val Ser Val Gly Asn Thr Leu Tyr Tyr Val Asn Lys Gln Glu Gly
450 455 460
Lys Ser Leu Tyr Val Lys Gly Glu Pro Ile Ile Asn Phe Tyr Asp Pro
465 470 475 480
Leu Val Phe Pro Ser Asp Glu Phe Asp Ala Ser Ile Ser Gln Val Asn
485 490 495
Glu Lys Ile Asn Gln Ser Leu Ala Phe Ile Arg Lys Ser Asp Glu Leu
500 505 510
Leu Gly Ser Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr
515 520 525
Val Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Gly
530 535 540
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
545 550 555 560
Gly Ser Ala Ser Arg Glu Leu Val Val Ser Tyr Val Asn Val Asn Met
565 570 575
Gly Leu Lys Ile Arg Gln Leu Leu Trp Phe His Ile Ser Ala Leu Thr
580 585 590
Phe Gly Arg Glu Thr Val Leu Glu Tyr Leu Val Ser Phe Gly Val Trp
595 600 605
Ile Arg Thr Pro Pro Ala Ala Arg Pro Pro Asn Ala Pro Ile Leu Ser
610 615 620
Thr Leu Pro Glu Thr Thr Val Val Gly Gly Gly Gly Ser Gly Gly Gly
625 630 635 640
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Ala Ser Val
645 650 655
Glu Leu Leu Ser Phe Leu Pro Ser Asp Phe Phe Pro Ser Ile Arg Asp
660 665 670
Leu Leu Asp Thr Ala Ser Ala Leu Tyr Arg Glu Ala Leu Glu Ser Pro
675 680 685
Glu His Ser Ser Pro His His Thr Ala Leu Arg Gln Ala Ile Leu Cys
690 695 700
Trp Gly Glu Leu Met Asn Leu Ala Thr Trp Val Gly Ser Gly Gly Ser
705 710 715 720
Asp Tyr Lys Asp Asp Asp Asp Lys
725
<210> 57
<211> 2307
<212> ДНК
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-Fc
<400> 57
atggaacttc tcatcctgaa agccaatgcc attacgacca ttctgacagc cgtaaccttt 60
tgctttgcaa gtggtcagaa tatcactgag gaattctacc agagtacctg tagcgcagtt 120
tctaaggggt acttgtctgc gcttcgcact ggctggtaca cctcggttat taccattgag 180
ctgtccaaca tcaaagagaa caagtgcaac ggcaccgacg ccaaagtgaa actgattaaa 240
caggagctcg acaaatacaa aaatgctgtg acagagttgc agttgctcat gcagtctacc 300
cctgctacga acaaccgagc aaggcgagaa ctaccaagat tcatgaacta caccctgaat 360
aatgcgaaaa agactaacgt gaccctcagc aagaagagaa agaggcgttt tcttgggttt 420
ttgctgggag ttgggagcgc tatagccagc ggagttgctg tgtgtaaggt attgcaccta 480
gaaggggagg tgaataagat caagagtgcc ctgctgtcca caaacaaagc agtagtgtcg 540
cttagcaatg gggtgtccgt tctgaccttc aaggttctag atctcaaaaa ctacatcgat 600
aagcagcttc tgccaatcct caacaagcag tcttgcagta ttagcaatat cgagacggtg 660
atagagtttc aacagaagaa caatcgtctg ctcgaaatta cacgggagtt tagcgtcaac 720
gcaggagtga ctactcctgt aagcacctac atgttaacaa actccgagct gctatctctg 780
atcaatgaca tgccaattac aaacgaccag aaaaagttaa tgtcaaacaa tgtgcagata 840
gtcaggcaac agtcctattc cattatgtgc atcatcaagg aagaagttct ggcctatgtc 900
gtccaacttc ccttatatgg cgtcatagac acgccctgtt ggaaactgca caccagtcct 960
ttgtgcacta caaacactaa ggaggggtct aacatctgtc tgactaggac agatcgcggg 1020
tggtattgcg acaatgctgg ctcagtgagc tttttcccac aagccgaaac atgcaaagtc 1080
cagtcgaatc gggtgttttg tgacacaatg aactcactga ctcttccttc cgaagtgaac 1140
ttgtgcaatg tcgatatctt caatcccaaa tatgactgca agatcatgac aagtaagacc 1200
gatgtcagca gtagcgtcat tacctccctc ggtgctattg tgtcctgtta cggcaagacc 1260
aaatgtactg cttctaacaa aaatcgcggc attattaaga cattcagcaa cggatgtgac 1320
tatgtctcca ataaaggtgt agacacggtg tctgtgggta ataccctcta ctatgtgaat 1380
aagcaggaag gaaagtcact ctatgtgaaa ggagagccga tcatcaactt ctacgatccc 1440
ctggtgtttc ccagtgatga gttcgacgcc tctatcagcc aggtgaatga aaagatcaac 1500
caatccctgg ccttcatacg gaaatcagat gagctgttag gctcaggcta catacccgaa 1560
gcaccgagag atggtcaagc gtatgtgcgg aaggatggag agtgggtcct tctgtcaact 1620
ttcctggaca aaactcacac ttgtccgcct tgtcccgctc cagagctcct tggcggaccc 1680
tccgttttcc tgtttcctcc gaaacccaag gatacgctga tgatttcacg cacaccagaa 1740
gtgacatgtg tggtggtaga tgtgtcccat gaagatccgg aggtgaagtt taactggtat 1800
gtcgatggtg tggaggttca taacgctaag acgaaaccac gggaggagca gtacaattcc 1860
acctaccgtg tagtctctgt gctgaccgtt ttgcatcagg attggctgaa tggtaaagag 1920
tataagtgca aagtgtccaa caaggctctg ccagccccta tcgaaaagac catcagtaaa 1980
gccaaaggac agcctagaga accccaggtc tatacgctgc caccctctcg ggacgagctg 2040
accaagaatc aggtgtcact gacttgtctg gtgaagggct tctaccctag cgacattgcc 2100
gtcgaatggg agtctaatgg gcaacccgag aataactaca aaaccacacc gcccgtcttg 2160
gacagcgatg gcagtttctt cctgtactcc aaactgactg tggacaaaag ccggtggcag 2220
caaggcaatg tgttctcatg ctctgtcatg cacgaagccc tgcacaacca ttacacccaa 2280
aagtcactga gcctgagccc tggaaag 2307
<210> 58
<211> 2307
<212> ДНК
<213> Искусственная последовательность
<220>
<223> FH_82-Fc
<400> 58
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agccgagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcgag 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaata ccgacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gctcaggcta catacccgaa 1560
gcaccgagag atggtcaagc gtatgtgcgg aaggatggag agtgggtcct tctgtcaact 1620
ttcctggaca aaactcacac ttgtccgcct tgtcccgctc cagagctcct tggcggaccc 1680
tccgttttcc tgtttcctcc gaaacccaag gatacgctga tgatttcacg cacaccagaa 1740
gtgacatgtg tggtggtaga tgtgtcccat gaagatccgg aggtgaagtt taactggtat 1800
gtcgatggtg tggaggttca taacgctaag acgaaaccac gggaggagca gtacaattcc 1860
acctaccgtg tagtctctgt gctgaccgtt ttgcatcagg attggctgaa tggtaaagag 1920
tataagtgca aagtgtccaa caaggctctg ccagccccta tcgaaaagac catcagtaaa 1980
gccaaaggac agcctagaga accccaggtc tatacgctgc caccctctcg ggacgagctg 2040
accaagaatc aggtgtcact gacttgtctg gtgaagggct tctaccctag cgacattgcc 2100
gtcgaatggg agtctaatgg gcaacccgag aataactaca aaaccacacc gcccgtcttg 2160
gacagcgatg gcagtttctt cctgtactcc aaactgactg tggacaaaag ccggtggcag 2220
caaggcaatg tgttctcatg ctctgtcatg cacgaagccc tgcacaacca ttacacccaa 2280
aagtcactga gcctgagccc tggaaag 2307
<210> 59
<211> 2307
<212> ДНК
<213> Искусственная последовательность
<220>
<223> FH_85-Fc
<400> 59
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agccgagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcatg 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaata ccgacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gctcaggcta catacccgaa 1560
gcaccgagag atggtcaagc gtatgtgcgg aaggatggag agtgggtcct tctgtcaact 1620
ttcctggaca aaactcacac ttgtccgcct tgtcccgctc cagagctcct tggcggaccc 1680
tccgttttcc tgtttcctcc gaaacccaag gatacgctga tgatttcacg cacaccagaa 1740
gtgacatgtg tggtggtaga tgtgtcccat gaagatccgg aggtgaagtt taactggtat 1800
gtcgatggtg tggaggttca taacgctaag acgaaaccac gggaggagca gtacaattcc 1860
acctaccgtg tagtctctgt gctgaccgtt ttgcatcagg attggctgaa tggtaaagag 1920
tataagtgca aagtgtccaa caaggctctg ccagccccta tcgaaaagac catcagtaaa 1980
gccaaaggac agcctagaga accccaggtc tatacgctgc caccctctcg ggacgagctg 2040
accaagaatc aggtgtcact gacttgtctg gtgaagggct tctaccctag cgacattgcc 2100
gtcgaatggg agtctaatgg gcaacccgag aataactaca aaaccacacc gcccgtcttg 2160
gacagcgatg gcagtttctt cctgtactcc aaactgactg tggacaaaag ccggtggcag 2220
caaggcaatg tgttctcatg ctctgtcatg cacgaagccc tgcacaacca ttacacccaa 2280
aagtcactga gcctgagccc tggaaag 2307
<210> 60
<211> 2367
<212> ДНК
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-4GS-Fc
<400> 60
atggaacttc tcatcctgaa agccaatgcc attacgacca ttctgacagc cgtaaccttt 60
tgctttgcaa gtggtcagaa tatcactgag gaattctacc agagtacctg tagcgcagtt 120
tctaaggggt acttgtctgc gcttcgcact ggctggtaca cctcggttat taccattgag 180
ctgtccaaca tcaaagagaa caagtgcaac ggcaccgacg ccaaagtgaa actgattaaa 240
caggagctcg acaaatacaa aaatgctgtg acagagttgc agttgctcat gcagtctacc 300
cctgctacga acaaccgagc aaggcgagaa ctaccaagat tcatgaacta caccctgaat 360
aatgcgaaaa agactaacgt gaccctcagc aagaagagaa agaggcgttt tcttgggttt 420
ttgctgggag ttgggagcgc tatagccagc ggagttgctg tgtgtaaggt attgcaccta 480
gaaggggagg tgaataagat caagagtgcc ctgctgtcca caaacaaagc agtagtgtcg 540
cttagcaatg gggtgtccgt tctgaccttc aaggttctag atctcaaaaa ctacatcgat 600
aagcagcttc tgccaatcct caacaagcag tcttgcagta ttagcaatat cgagacggtg 660
atagagtttc aacagaagaa caatcgtctg ctcgaaatta cacgggagtt tagcgtcaac 720
gcaggagtga ctactcctgt aagcacctac atgttaacaa actccgagct gctatctctg 780
atcaatgaca tgccaattac aaacgaccag aaaaagttaa tgtcaaacaa tgtgcagata 840
gtcaggcaac agtcctattc cattatgtgc atcatcaagg aagaagttct ggcctatgtc 900
gtccaacttc ccttatatgg cgtcatagac acgccctgtt ggaaactgca caccagtcct 960
ttgtgcacta caaacactaa ggaggggtct aacatctgtc tgactaggac agatcgcggg 1020
tggtattgcg acaatgctgg ctcagtgagc tttttcccac aagccgaaac atgcaaagtc 1080
cagtcgaatc gggtgttttg tgacacaatg aactcactga ctcttccttc cgaagtgaac 1140
ttgtgcaatg tcgatatctt caatcccaaa tatgactgca agatcatgac aagtaagacc 1200
gatgtcagca gtagcgtcat tacctccctc ggtgctattg tgtcctgtta cggcaagacc 1260
aaatgtactg cttctaacaa aaatcgcggc attattaaga cattcagcaa cggatgtgac 1320
tatgtctcca ataaaggtgt agacacggtg tctgtgggta ataccctcta ctatgtgaat 1380
aagcaggaag gaaagtcact ctatgtgaaa ggagagccga tcatcaactt ctacgatccc 1440
ctggtgtttc ccagtgatga gttcgacgcc tctatcagcc aggtgaatga aaagatcaac 1500
caatccctgg ccttcatacg gaaatcagat gagctgttag gctcaggcta catacccgaa 1560
gcaccgagag atggtcaagc gtatgtgcgg aaggatggag agtgggtcct tctgtcaact 1620
ttcctgggcg ggggtgggag cggaggcgga ggttcagggg gtggagggtc aggcggagga 1680
ggcagcgaca aaactcacac ttgtccgcct tgtcccgctc cagagctcct tggcggaccc 1740
tccgttttcc tgtttcctcc gaaacccaag gatacgctga tgatttcacg cacaccagaa 1800
gtgacatgtg tggtggtaga tgtgtcccat gaagatccgg aggtgaagtt taactggtat 1860
gtcgatggtg tggaggttca taacgctaag acgaaaccac gggaggagca gtacaattcc 1920
acctaccgtg tagtctctgt gctgaccgtt ttgcatcagg attggctgaa tggtaaagag 1980
tataagtgca aagtgtccaa caaggctctg ccagccccta tcgaaaagac catcagtaaa 2040
gccaaaggac agcctagaga accccaggtc tatacgctgc caccctctcg ggacgagctg 2100
accaagaatc aggtgtcact gacttgtctg gtgaagggct tctaccctag cgacattgcc 2160
gtcgaatggg agtctaatgg gcaacccgag aataactaca aaaccacacc gcccgtcttg 2220
gacagcgatg gcagtttctt cctgtactcc aaactgactg tggacaaaag ccggtggcag 2280
caaggcaatg tgttctcatg ctctgtcatg cacgaagccc tgcacaacca ttacacccaa 2340
aagtcactga gcctgagccc tggaaag 2367
<210> 61
<211> 2337
<212> ДНК
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-2GS-Fc
<400> 61
atggaacttc tcatcctgaa agccaatgcc attacgacca ttctgacagc cgtaaccttt 60
tgctttgcaa gtggtcagaa tatcactgag gaattctacc agagtacctg tagcgcagtt 120
tctaaggggt acttgtctgc gcttcgcact ggctggtaca cctcggttat taccattgag 180
ctgtccaaca tcaaagagaa caagtgcaac ggcaccgacg ccaaagtgaa actgattaaa 240
caggagctcg acaaatacaa aaatgctgtg acagagttgc agttgctcat gcagtctacc 300
cctgctacga acaaccgagc aaggcgagaa ctaccaagat tcatgaacta caccctgaat 360
aatgcgaaaa agactaacgt gaccctcagc aagaagagaa agaggcgttt tcttgggttt 420
ttgctgggag ttgggagcgc tatagccagc ggagttgctg tgtgtaaggt attgcaccta 480
gaaggggagg tgaataagat caagagtgcc ctgctgtcca caaacaaagc agtagtgtcg 540
cttagcaatg gggtgtccgt tctgaccttc aaggttctag atctcaaaaa ctacatcgat 600
aagcagcttc tgccaatcct caacaagcag tcttgcagta ttagcaatat cgagacggtg 660
atagagtttc aacagaagaa caatcgtctg ctcgaaatta cacgggagtt tagcgtcaac 720
gcaggagtga ctactcctgt aagcacctac atgttaacaa actccgagct gctatctctg 780
atcaatgaca tgccaattac aaacgaccag aaaaagttaa tgtcaaacaa tgtgcagata 840
gtcaggcaac agtcctattc cattatgtgc atcatcaagg aagaagttct ggcctatgtc 900
gtccaacttc ccttatatgg cgtcatagac acgccctgtt ggaaactgca caccagtcct 960
ttgtgcacta caaacactaa ggaggggtct aacatctgtc tgactaggac agatcgcggg 1020
tggtattgcg acaatgctgg ctcagtgagc tttttcccac aagccgaaac atgcaaagtc 1080
cagtcgaatc gggtgttttg tgacacaatg aactcactga ctcttccttc cgaagtgaac 1140
ttgtgcaatg tcgatatctt caatcccaaa tatgactgca agatcatgac aagtaagacc 1200
gatgtcagca gtagcgtcat tacctccctc ggtgctattg tgtcctgtta cggcaagacc 1260
aaatgtactg cttctaacaa aaatcgcggc attattaaga cattcagcaa cggatgtgac 1320
tatgtctcca ataaaggtgt agacacggtg tctgtgggta ataccctcta ctatgtgaat 1380
aagcaggaag gaaagtcact ctatgtgaaa ggagagccga tcatcaactt ctacgatccc 1440
ctggtgtttc ccagtgatga gttcgacgcc tctatcagcc aggtgaatga aaagatcaac 1500
caatccctgg ccttcatacg gaaatcagat gagctgttag gctcaggcta catacccgaa 1560
gcaccgagag atggtcaagc gtatgtgcgg aaggatggag agtgggtcct tctgtcaact 1620
ttcctggggg gtggagggtc aggcggagga ggcagcgaca aaactcacac ttgtccgcct 1680
tgtcccgctc cagagctcct tggcggaccc tccgttttcc tgtttcctcc gaaacccaag 1740
gatacgctga tgatttcacg cacaccagaa gtgacatgtg tggtggtaga tgtgtcccat 1800
gaagatccgg aggtgaagtt taactggtat gtcgatggtg tggaggttca taacgctaag 1860
acgaaaccac gggaggagca gtacaattcc acctaccgtg tagtctctgt gctgaccgtt 1920
ttgcatcagg attggctgaa tggtaaagag tataagtgca aagtgtccaa caaggctctg 1980
ccagccccta tcgaaaagac catcagtaaa gccaaaggac agcctagaga accccaggtc 2040
tatacgctgc caccctctcg ggacgagctg accaagaatc aggtgtcact gacttgtctg 2100
gtgaagggct tctaccctag cgacattgcc gtcgaatggg agtctaatgg gcaacccgag 2160
aataactaca aaaccacacc gcccgtcttg gacagcgatg gcagtttctt cctgtactcc 2220
aaactgactg tggacaaaag ccggtggcag caaggcaatg tgttctcatg ctctgtcatg 2280
cacgaagccc tgcacaacca ttacacccaa aagtcactga gcctgagccc tggaaag 2337
<210> 62
<211> 2322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-1GS-Fc
<400> 62
atggaacttc tcatcctgaa agccaatgcc attacgacca ttctgacagc cgtaaccttt 60
tgctttgcaa gtggtcagaa tatcactgag gaattctacc agagtacctg tagcgcagtt 120
tctaaggggt acttgtctgc gcttcgcact ggctggtaca cctcggttat taccattgag 180
ctgtccaaca tcaaagagaa caagtgcaac ggcaccgacg ccaaagtgaa actgattaaa 240
caggagctcg acaaatacaa aaatgctgtg acagagttgc agttgctcat gcagtctacc 300
cctgctacga acaaccgagc aaggcgagaa ctaccaagat tcatgaacta caccctgaat 360
aatgcgaaaa agactaacgt gaccctcagc aagaagagaa agaggcgttt tcttgggttt 420
ttgctgggag ttgggagcgc tatagccagc ggagttgctg tgtgtaaggt attgcaccta 480
gaaggggagg tgaataagat caagagtgcc ctgctgtcca caaacaaagc agtagtgtcg 540
cttagcaatg gggtgtccgt tctgaccttc aaggttctag atctcaaaaa ctacatcgat 600
aagcagcttc tgccaatcct caacaagcag tcttgcagta ttagcaatat cgagacggtg 660
atagagtttc aacagaagaa caatcgtctg ctcgaaatta cacgggagtt tagcgtcaac 720
gcaggagtga ctactcctgt aagcacctac atgttaacaa actccgagct gctatctctg 780
atcaatgaca tgccaattac aaacgaccag aaaaagttaa tgtcaaacaa tgtgcagata 840
gtcaggcaac agtcctattc cattatgtgc atcatcaagg aagaagttct ggcctatgtc 900
gtccaacttc ccttatatgg cgtcatagac acgccctgtt ggaaactgca caccagtcct 960
ttgtgcacta caaacactaa ggaggggtct aacatctgtc tgactaggac agatcgcggg 1020
tggtattgcg acaatgctgg ctcagtgagc tttttcccac aagccgaaac atgcaaagtc 1080
cagtcgaatc gggtgttttg tgacacaatg aactcactga ctcttccttc cgaagtgaac 1140
ttgtgcaatg tcgatatctt caatcccaaa tatgactgca agatcatgac aagtaagacc 1200
gatgtcagca gtagcgtcat tacctccctc ggtgctattg tgtcctgtta cggcaagacc 1260
aaatgtactg cttctaacaa aaatcgcggc attattaaga cattcagcaa cggatgtgac 1320
tatgtctcca ataaaggtgt agacacggtg tctgtgggta ataccctcta ctatgtgaat 1380
aagcaggaag gaaagtcact ctatgtgaaa ggagagccga tcatcaactt ctacgatccc 1440
ctggtgtttc ccagtgatga gttcgacgcc tctatcagcc aggtgaatga aaagatcaac 1500
caatccctgg ccttcatacg gaaatcagat gagctgttag gctcaggcta catacccgaa 1560
gcaccgagag atggtcaagc gtatgtgcgg aaggatggag agtgggtcct tctgtcaact 1620
ttcctgggcg gaggaggcag cgacaaaact cacacttgtc cgccttgtcc cgctccagag 1680
ctccttggcg gaccctccgt tttcctgttt cctccgaaac ccaaggatac gctgatgatt 1740
tcacgcacac cagaagtgac atgtgtggtg gtagatgtgt cccatgaaga tccggaggtg 1800
aagtttaact ggtatgtcga tggtgtggag gttcataacg ctaagacgaa accacgggag 1860
gagcagtaca attccaccta ccgtgtagtc tctgtgctga ccgttttgca tcaggattgg 1920
ctgaatggta aagagtataa gtgcaaagtg tccaacaagg ctctgccagc ccctatcgaa 1980
aagaccatca gtaaagccaa aggacagcct agagaacccc aggtctatac gctgccaccc 2040
tctcgggacg agctgaccaa gaatcaggtg tcactgactt gtctggtgaa gggcttctac 2100
cctagcgaca ttgccgtcga atgggagtct aatgggcaac ccgagaataa ctacaaaacc 2160
acaccgcccg tcttggacag cgatggcagt ttcttcctgt actccaaact gactgtggac 2220
aaaagccggt ggcagcaagg caatgtgttc tcatgctctg tcatgcacga agccctgcac 2280
aaccattaca cccaaaagtc actgagcctg agccctggaa ag 2322
<210> 63
<211> 2367
<212> ДНК
<213> Искусственная последовательность
<220>
<223> FH_85-4GS-Fc
<400> 63
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agccgagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcatg 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaata ccgacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gctcaggcta catacccgaa 1560
gcaccgagag atggtcaagc gtatgtgcgg aaggatggag agtgggtcct tctgtcaact 1620
ttcctgggcg ggggtgggag cggaggcgga ggttcagggg gtggagggtc aggcggagga 1680
ggcagcgaca aaactcacac ttgtccgcct tgtcccgctc cagagctcct tggcggaccc 1740
tccgttttcc tgtttcctcc gaaacccaag gatacgctga tgatttcacg cacaccagaa 1800
gtgacatgtg tggtggtaga tgtgtcccat gaagatccgg aggtgaagtt taactggtat 1860
gtcgatggtg tggaggttca taacgctaag acgaaaccac gggaggagca gtacaattcc 1920
acctaccgtg tagtctctgt gctgaccgtt ttgcatcagg attggctgaa tggtaaagag 1980
tataagtgca aagtgtccaa caaggctctg ccagccccta tcgaaaagac catcagtaaa 2040
gccaaaggac agcctagaga accccaggtc tatacgctgc caccctctcg ggacgagctg 2100
accaagaatc aggtgtcact gacttgtctg gtgaagggct tctaccctag cgacattgcc 2160
gtcgaatggg agtctaatgg gcaacccgag aataactaca aaaccacacc gcccgtcttg 2220
gacagcgatg gcagtttctt cctgtactcc aaactgactg tggacaaaag ccggtggcag 2280
caaggcaatg tgttctcatg ctctgtcatg cacgaagccc tgcacaacca ttacacccaa 2340
aagtcactga gcctgagccc tggaaag 2367
<210> 64
<211> 2337
<212> ДНК
<213> Искусственная последовательность
<220>
<223> FH_85-2GS-Fc
<400> 64
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agccgagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcatg 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaata ccgacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gctcaggcta catacccgaa 1560
gcaccgagag atggtcaagc gtatgtgcgg aaggatggag agtgggtcct tctgtcaact 1620
ttcctggggg gtggagggtc aggcggagga ggcagcgaca aaactcacac ttgtccgcct 1680
tgtcccgctc cagagctcct tggcggaccc tccgttttcc tgtttcctcc gaaacccaag 1740
gatacgctga tgatttcacg cacaccagaa gtgacatgtg tggtggtaga tgtgtcccat 1800
gaagatccgg aggtgaagtt taactggtat gtcgatggtg tggaggttca taacgctaag 1860
acgaaaccac gggaggagca gtacaattcc acctaccgtg tagtctctgt gctgaccgtt 1920
ttgcatcagg attggctgaa tggtaaagag tataagtgca aagtgtccaa caaggctctg 1980
ccagccccta tcgaaaagac catcagtaaa gccaaaggac agcctagaga accccaggtc 2040
tatacgctgc caccctctcg ggacgagctg accaagaatc aggtgtcact gacttgtctg 2100
gtgaagggct tctaccctag cgacattgcc gtcgaatggg agtctaatgg gcaacccgag 2160
aataactaca aaaccacacc gcccgtcttg gacagcgatg gcagtttctt cctgtactcc 2220
aaactgactg tggacaaaag ccggtggcag caaggcaatg tgttctcatg ctctgtcatg 2280
cacgaagccc tgcacaacca ttacacccaa aagtcactga gcctgagccc tggaaag 2337
<210> 65
<211> 2322
<212> ДНК
<213> Искусственная последовательность
<220>
<223> FH_85-1GS-Fc
<400> 65
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agccgagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcatg 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaata ccgacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gctcaggcta catacccgaa 1560
gcaccgagag atggtcaagc gtatgtgcgg aaggatggag agtgggtcct tctgtcaact 1620
ttcctgggcg gaggaggcag cgacaaaact cacacttgtc cgccttgtcc cgctccagag 1680
ctccttggcg gaccctccgt tttcctgttt cctccgaaac ccaaggatac gctgatgatt 1740
tcacgcacac cagaagtgac atgtgtggtg gtagatgtgt cccatgaaga tccggaggtg 1800
aagtttaact ggtatgtcga tggtgtggag gttcataacg ctaagacgaa accacgggag 1860
gagcagtaca attccaccta ccgtgtagtc tctgtgctga ccgttttgca tcaggattgg 1920
ctgaatggta aagagtataa gtgcaaagtg tccaacaagg ctctgccagc ccctatcgaa 1980
aagaccatca gtaaagccaa aggacagcct agagaacccc aggtctatac gctgccaccc 2040
tctcgggacg agctgaccaa gaatcaggtg tcactgactt gtctggtgaa gggcttctac 2100
cctagcgaca ttgccgtcga atgggagtct aatgggcaac ccgagaataa ctacaaaacc 2160
acaccgcccg tcttggacag cgatggcagt ttcttcctgt actccaaact gactgtggac 2220
aaaagccggt ggcagcaagg caatgtgttc tcatgctctg tcatgcacga agccctgcac 2280
aaccattaca cccaaaagtc actgagcctg agccctggaa ag 2322
<210> 66
<211> 1713
<212> ДНК
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-CC02
<400> 66
atggaacttc tcatcctgaa agccaatgcc attacgacca ttctgacagc cgtaaccttt 60
tgctttgcaa gtggtcagaa tatcactgag gaattctacc agagtacctg tagcgcagtt 120
tctaaggggt acttgtctgc gcttcgcact ggctggtaca cctcggttat taccattgag 180
ctgtccaaca tcaaagagaa caagtgcaac ggcaccgacg ccaaagtgaa actgattaaa 240
caggagctcg acaaatacaa aaatgctgtg acagagttgc agttgctcat gcagtctacc 300
cctgctacga acaaccgagc aaggcgagaa ctaccaagat tcatgaacta caccctgaat 360
aatgcgaaaa agactaacgt gaccctcagc aagaagagaa agaggcgttt tcttgggttt 420
ttgctgggag ttgggagcgc tatagccagc ggagttgctg tgtgtaaggt attgcaccta 480
gaaggggagg tgaataagat caagagtgcc ctgctgtcca caaacaaagc agtagtgtcg 540
cttagcaatg gggtgtccgt tctgaccttc aaggttctag atctcaaaaa ctacatcgat 600
aagcagcttc tgccaatcct caacaagcag tcttgcagta ttagcaatat cgagacggtg 660
atagagtttc aacagaagaa caatcgtctg ctcgaaatta cacgggagtt tagcgtcaac 720
gcaggagtga ctactcctgt aagcacctac atgttaacaa actccgagct gctatctctg 780
atcaatgaca tgccaattac aaacgaccag aaaaagttaa tgtcaaacaa tgtgcagata 840
gtcaggcaac agtcctattc cattatgtgc atcatcaagg aagaagttct ggcctatgtc 900
gtccaacttc ccttatatgg cgtcatagac acgccctgtt ggaaactgca caccagtcct 960
ttgtgcacta caaacactaa ggaggggtct aacatctgtc tgactaggac agatcgcggg 1020
tggtattgcg acaatgctgg ctcagtgagc tttttcccac aagccgaaac atgcaaagtc 1080
cagtcgaatc gggtgttttg tgacacaatg aactcactga ctcttccttc cgaagtgaac 1140
ttgtgcaatg tcgatatctt caatcccaaa tatgactgca agatcatgac aagtaagacc 1200
gatgtcagca gtagcgtcat tacctccctc ggtgctattg tgtcctgtta cggcaagacc 1260
aaatgtactg cttctaacaa aaatcgcggc attattaaga cattcagcaa cggatgtgac 1320
tatgtctcca ataaaggtgt agacacggtg tctgtgggta ataccctcta ctatgtgaat 1380
aagcaggaag gaaagtcact ctatgtgaaa ggagagccga tcatcaactt ctacgatccc 1440
ctggtgtttc ccagtgatga gttcgacgcc tctatcagcc aggtgaatga aaagatcaac 1500
caatccctgg ccttcatacg gaaatcagat gagctgttag ggtctggcta catccctgaa 1560
gcacccagag atggtcaggc ctatgtgagg aaagatggcg aatgggtcct gctgtccacc 1620
ttcctgatag cgttggccct ggagaaaatc gcccttgcac tggagaagat tgccctcgcc 1680
ttggagaagg actacaagga cgatgacgac aaa 1713
<210> 67
<211> 1713
<212> ДНК
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-CC03
<400> 67
atggaacttc tcatcctgaa agccaatgcc attacgacca ttctgacagc cgtaaccttt 60
tgctttgcaa gtggtcagaa tatcactgag gaattctacc agagtacctg tagcgcagtt 120
tctaaggggt acttgtctgc gcttcgcact ggctggtaca cctcggttat taccattgag 180
ctgtccaaca tcaaagagaa caagtgcaac ggcaccgacg ccaaagtgaa actgattaaa 240
caggagctcg acaaatacaa aaatgctgtg acagagttgc agttgctcat gcagtctacc 300
cctgctacga acaaccgagc aaggcgagaa ctaccaagat tcatgaacta caccctgaat 360
aatgcgaaaa agactaacgt gaccctcagc aagaagagaa agaggcgttt tcttgggttt 420
ttgctgggag ttgggagcgc tatagccagc ggagttgctg tgtgtaaggt attgcaccta 480
gaaggggagg tgaataagat caagagtgcc ctgctgtcca caaacaaagc agtagtgtcg 540
cttagcaatg gggtgtccgt tctgaccttc aaggttctag atctcaaaaa ctacatcgat 600
aagcagcttc tgccaatcct caacaagcag tcttgcagta ttagcaatat cgagacggtg 660
atagagtttc aacagaagaa caatcgtctg ctcgaaatta cacgggagtt tagcgtcaac 720
gcaggagtga ctactcctgt aagcacctac atgttaacaa actccgagct gctatctctg 780
atcaatgaca tgccaattac aaacgaccag aaaaagttaa tgtcaaacaa tgtgcagata 840
gtcaggcaac agtcctattc cattatgtgc atcatcaagg aagaagttct ggcctatgtc 900
gtccaacttc ccttatatgg cgtcatagac acgccctgtt ggaaactgca caccagtcct 960
ttgtgcacta caaacactaa ggaggggtct aacatctgtc tgactaggac agatcgcggg 1020
tggtattgcg acaatgctgg ctcagtgagc tttttcccac aagccgaaac atgcaaagtc 1080
cagtcgaatc gggtgttttg tgacacaatg aactcactga ctcttccttc cgaagtgaac 1140
ttgtgcaatg tcgatatctt caatcccaaa tatgactgca agatcatgac aagtaagacc 1200
gatgtcagca gtagcgtcat tacctccctc ggtgctattg tgtcctgtta cggcaagacc 1260
aaatgtactg cttctaacaa aaatcgcggc attattaaga cattcagcaa cggatgtgac 1320
tatgtctcca ataaaggtgt agacacggtg tctgtgggta ataccctcta ctatgtgaat 1380
aagcaggaag gaaagtcact ctatgtgaaa ggagagccga tcatcaactt ctacgatccc 1440
ctggtgtttc ccagtgatga gttcgacgcc tctatcagcc aggtgaatga aaagatcaac 1500
caatccctgg ccttcatacg gaaatcagat gagctgttag gatctggcta cattcccgaa 1560
gctcctagag atggccaggc ctatgtgagg aaagatgggg agtgggtgtt gctgagcacc 1620
ttcctcatca aactggagct ggagaagatt aagctggaac tggagaaaat caagctcgaa 1680
cttgagaagg actacaagga cgatgacgac aaa 1713
<210> 68
<211> 1713
<212> ДНК
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-CC07
<400> 68
atggaacttc tcatcctgaa agccaatgcc attacgacca ttctgacagc cgtaaccttt 60
tgctttgcaa gtggtcagaa tatcactgag gaattctacc agagtacctg tagcgcagtt 120
tctaaggggt acttgtctgc gcttcgcact ggctggtaca cctcggttat taccattgag 180
ctgtccaaca tcaaagagaa caagtgcaac ggcaccgacg ccaaagtgaa actgattaaa 240
caggagctcg acaaatacaa aaatgctgtg acagagttgc agttgctcat gcagtctacc 300
cctgctacga acaaccgagc aaggcgagaa ctaccaagat tcatgaacta caccctgaat 360
aatgcgaaaa agactaacgt gaccctcagc aagaagagaa agaggcgttt tcttgggttt 420
ttgctgggag ttgggagcgc tatagccagc ggagttgctg tgtgtaaggt attgcaccta 480
gaaggggagg tgaataagat caagagtgcc ctgctgtcca caaacaaagc agtagtgtcg 540
cttagcaatg gggtgtccgt tctgaccttc aaggttctag atctcaaaaa ctacatcgat 600
aagcagcttc tgccaatcct caacaagcag tcttgcagta ttagcaatat cgagacggtg 660
atagagtttc aacagaagaa caatcgtctg ctcgaaatta cacgggagtt tagcgtcaac 720
gcaggagtga ctactcctgt aagcacctac atgttaacaa actccgagct gctatctctg 780
atcaatgaca tgccaattac aaacgaccag aaaaagttaa tgtcaaacaa tgtgcagata 840
gtcaggcaac agtcctattc cattatgtgc atcatcaagg aagaagttct ggcctatgtc 900
gtccaacttc ccttatatgg cgtcatagac acgccctgtt ggaaactgca caccagtcct 960
ttgtgcacta caaacactaa ggaggggtct aacatctgtc tgactaggac agatcgcggg 1020
tggtattgcg acaatgctgg ctcagtgagc tttttcccac aagccgaaac atgcaaagtc 1080
cagtcgaatc gggtgttttg tgacacaatg aactcactga ctcttccttc cgaagtgaac 1140
ttgtgcaatg tcgatatctt caatcccaaa tatgactgca agatcatgac aagtaagacc 1200
gatgtcagca gtagcgtcat tacctccctc ggtgctattg tgtcctgtta cggcaagacc 1260
aaatgtactg cttctaacaa aaatcgcggc attattaaga cattcagcaa cggatgtgac 1320
tatgtctcca ataaaggtgt agacacggtg tctgtgggta ataccctcta ctatgtgaat 1380
aagcaggaag gaaagtcact ctatgtgaaa ggagagccga tcatcaactt ctacgatccc 1440
ctggtgtttc ccagtgatga gttcgacgcc tctatcagcc aggtgaatga aaagatcaac 1500
caatccctgg ccttcatacg gaaatcagat gagctgttag gttccgggta catacccgaa 1560
gctcctagag atggacaggc ctatgtgagg aaagatggcg aatgggtctt actgagcacc 1620
ttcttgctcc atctgcacat tgagaagcta cacctccata tcgagaaact gcatcttcac 1680
atcgagaagg actacaagga cgatgacgac aaa 1713
<210> 69
<211> 1713
<212> ДНК
<213> Искусственная последовательность
<220>
<223> F_DS-Cav1-CC08
<400> 69
atggaacttc tcatcctgaa agccaatgcc attacgacca ttctgacagc cgtaaccttt 60
tgctttgcaa gtggtcagaa tatcactgag gaattctacc agagtacctg tagcgcagtt 120
tctaaggggt acttgtctgc gcttcgcact ggctggtaca cctcggttat taccattgag 180
ctgtccaaca tcaaagagaa caagtgcaac ggcaccgacg ccaaagtgaa actgattaaa 240
caggagctcg acaaatacaa aaatgctgtg acagagttgc agttgctcat gcagtctacc 300
cctgctacga acaaccgagc aaggcgagaa ctaccaagat tcatgaacta caccctgaat 360
aatgcgaaaa agactaacgt gaccctcagc aagaagagaa agaggcgttt tcttgggttt 420
ttgctgggag ttgggagcgc tatagccagc ggagttgctg tgtgtaaggt attgcaccta 480
gaaggggagg tgaataagat caagagtgcc ctgctgtcca caaacaaagc agtagtgtcg 540
cttagcaatg gggtgtccgt tctgaccttc aaggttctag atctcaaaaa ctacatcgat 600
aagcagcttc tgccaatcct caacaagcag tcttgcagta ttagcaatat cgagacggtg 660
atagagtttc aacagaagaa caatcgtctg ctcgaaatta cacgggagtt tagcgtcaac 720
gcaggagtga ctactcctgt aagcacctac atgttaacaa actccgagct gctatctctg 780
atcaatgaca tgccaattac aaacgaccag aaaaagttaa tgtcaaacaa tgtgcagata 840
gtcaggcaac agtcctattc cattatgtgc atcatcaagg aagaagttct ggcctatgtc 900
gtccaacttc ccttatatgg cgtcatagac acgccctgtt ggaaactgca caccagtcct 960
ttgtgcacta caaacactaa ggaggggtct aacatctgtc tgactaggac agatcgcggg 1020
tggtattgcg acaatgctgg ctcagtgagc tttttcccac aagccgaaac atgcaaagtc 1080
cagtcgaatc gggtgttttg tgacacaatg aactcactga ctcttccttc cgaagtgaac 1140
ttgtgcaatg tcgatatctt caatcccaaa tatgactgca agatcatgac aagtaagacc 1200
gatgtcagca gtagcgtcat tacctccctc ggtgctattg tgtcctgtta cggcaagacc 1260
aaatgtactg cttctaacaa aaatcgcggc attattaaga cattcagcaa cggatgtgac 1320
tatgtctcca ataaaggtgt agacacggtg tctgtgggta ataccctcta ctatgtgaat 1380
aagcaggaag gaaagtcact ctatgtgaaa ggagagccga tcatcaactt ctacgatccc 1440
ctggtgtttc ccagtgatga gttcgacgcc tctatcagcc aggtgaatga aaagatcaac 1500
caatccctgg ccttcatacg gaaatcagat gagctgttag gaagcggcta cattcccgaa 1560
gctcctagag atggtcaggc ctatgtgagg aaagatgggg aatgggtctt actgtccacc 1620
ttccttctca agttggagat ccaccatctg aagctagaga tccatcacct caaactggag 1680
atacaccatg actacaagga cgatgacgac aaa 1713
<210> 70
<211> 1713
<212> ДНК
<213> Искусственная последовательность
<220>
<223> FH_82-CC07
<400> 70
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agccgagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcgag 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaata ccgacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gatcaggcta cattcccgaa 1560
gctccaaggg acggacaagc atacgtacga aaagatggcg aatgggtctt actgagcacc 1620
ttcttgctcc atctgcacat tgagaagcta cacctccata tcgagaaact gcatcttcac 1680
atcgagaagg actacaagga cgatgacgac aaa 1713
<210> 71
<211> 1713
<212> ДНК
<213> Искусственная последовательность
<220>
<223> FH_85-CC07
<400> 71
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agccgagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcatg 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaata ccgacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gatcaggcta cattcccgaa 1560
gctccaaggg acggacaagc atacgtacga aaagatggcg aatgggtctt actgagcacc 1620
ttcttgctcc atctgcacat tgagaagcta cacctccata tcgagaaact gcatcttcac 1680
atcgagaagg actacaagga cgatgacgac aaa 1713
<210> 72
<211> 2184
<212> ДНК
<213> Искусственная последовательность
<220>
<223> F_DC-Cav1-HBcCN
<400> 72
atggaacttc tcatcctgaa agccaatgcc attacgacca ttctgacagc cgtaaccttt 60
tgctttgcaa gtggtcagaa tatcactgag gaattctacc agagtacctg tagcgcagtt 120
tctaaggggt acttgtctgc gcttcgcact ggctggtaca cctcggttat taccattgag 180
ctgtccaaca tcaaagagaa caagtgcaac ggcaccgacg ccaaagtgaa actgattaaa 240
caggagctcg acaaatacaa aaatgctgtg acagagttgc agttgctcat gcagtctacc 300
cctgctacga acaaccgagc aaggcgagaa ctaccaagat tcatgaacta caccctgaat 360
aatgcgaaaa agactaacgt gaccctcagc aagaagagaa agaggcgttt tcttgggttt 420
ttgctgggag ttgggagcgc tatagccagc ggagttgctg tgtgtaaggt attgcaccta 480
gaaggggagg tgaataagat caagagtgcc ctgctgtcca caaacaaagc agtagtgtcg 540
cttagcaatg gggtgtccgt tctgaccttc aaggttctag atctcaaaaa ctacatcgat 600
aagcagcttc tgccaatcct caacaagcag tcttgcagta ttagcaatat cgagacggtg 660
atagagtttc aacagaagaa caatcgtctg ctcgaaatta cacgggagtt tagcgtcaac 720
gcaggagtga ctactcctgt aagcacctac atgttaacaa actccgagct gctatctctg 780
atcaatgaca tgccaattac aaacgaccag aaaaagttaa tgtcaaacaa tgtgcagata 840
gtcaggcaac agtcctattc cattatgtgc atcatcaagg aagaagttct ggcctatgtc 900
gtccaacttc ccttatatgg cgtcatagac acgccctgtt ggaaactgca caccagtcct 960
ttgtgcacta caaacactaa ggaggggtct aacatctgtc tgactaggac agatcgcggg 1020
tggtattgcg acaatgctgg ctcagtgagc tttttcccac aagccgaaac atgcaaagtc 1080
cagtcgaatc gggtgttttg tgacacaatg aactcactga ctcttccttc cgaagtgaac 1140
ttgtgcaatg tcgatatctt caatcccaaa tatgactgca agatcatgac aagtaagacc 1200
gatgtcagca gtagcgtcat tacctccctc ggtgctattg tgtcctgtta cggcaagacc 1260
aaatgtactg cttctaacaa aaatcgcggc attattaaga cattcagcaa cggatgtgac 1320
tatgtctcca ataaaggtgt agacacggtg tctgtgggta ataccctcta ctatgtgaat 1380
aagcaggaag gaaagtcact ctatgtgaaa ggagagccga tcatcaactt ctacgatccc 1440
ctggtgtttc ccagtgatga gttcgacgcc tctatcagcc aggtgaatga aaagatcaac 1500
caatccctgg ccttcatacg gaaatcagat gagctgttag gctcaggcta catacccgaa 1560
gcaccgagag atggtcaagc gtatgtgcgg aaggatggag agtgggtcct tctgtcaact 1620
ttcctgggcg ggggtgggag cggaggcgga ggttcagggg gtggagggtc aggcggagga 1680
ggcagcgctt ccagggagtt agtggtctcc tacgtcaacg tgaacatggg ccttaagatc 1740
cgtcagttgc tgtggttcca catttccgcc ctaacctttg gcagagagac tgtgctggag 1800
tacctggtgt catttggcgt gtggattcgc actcctccag cagcaagacc cccaaatgcg 1860
cctatcctgt ccacgttacc agagaccacc gttgttgggg gaggtggttc tggagggggc 1920
ggatctggcg gaggtggcag tgggggcggt ggaagtggcg ctagcgtcga gttgctcagc 1980
ttccttccct cggacttctt tccgtccatc agggatctgc tggacacagc cagtgctctc 2040
tatcgggaag cactggaatc tcccgaacac agctctcctc accatacagc ccttcgacaa 2100
gccatactct gctggggcga actgatgaat ctcgccacat gggtagggtc aggggggagc 2160
gactacaagg acgatgacga caaa 2184
<210> 73
<211> 2184
<212> ДНК
<213> Искусственная последовательность
<220>
<223> FH_85-HBcCN
<400> 73
atggaactgc tgatacttaa ggctaacgcg ataactacga tcctcaccgc cgtgaccttt 60
tgctttgcct ctggccaaaa cattactgag gaattctacc agtcaacgtg cagtgcagtg 120
agccgagggt atctgtccgc cctgagaacc gggtggtata cttccgtcat taccatcatg 180
ctgtccaaca taaaagagaa taagtgcaac ggcaccgatg ctaaagtgaa actgatcaaa 240
caggaactcg ataaatacaa gaatgcagtt acagagcttc agctcctgat gcagagcact 300
ccggccacca ataatagggc aaggagagaa ttgccacgat ttatgaatta cacactcaac 360
aacgcgaaga aaactaacgt gactctgtcc aagaaacgta agaataactt cttggggttc 420
tgtctgggtg taggtagcgc cattgcttct ggggtggccg tcagcaaagt gcttcacctg 480
gaaggagagg tgaacaagat caagtctgca ctgctgtcta caaacaaagc agtggtgagc 540
ctgtccaacg gagtatccgt tctggtggtc aaagtcctgg atctgaagaa ttatatcgac 600
aaacaactgc tccccattgt gaacaagcag agttgttcaa tcagcaacat agaaactgtg 660
attgagttcc aacagaagaa caataggctg ctcgaaatta ccagagagtt tagcgtcaat 720
gctggtgtca caaccccagt cagcacttac atgctgacta attccgagtt gcttagcctt 780
attaacgaca tgcctatcac caatgaccag aagaagctga tgagtaataa tgtgcagatt 840
gtgcgccagc agagttacag cattatgagt attatcaaag aggaggtatt ggcttatgtg 900
gttcagcttc cgctgtatgg ggtcatcgac acaccttgtt ggaagttgca taccagtccc 960
ctgtgtacga caaacaccaa ggaaggtagt aacatctgct tgacacgtac cgatcggggt 1020
tggtattgcg ataacgccgg gtctgttagt ttctttcctc aagccgagac atgcaaagtc 1080
cagagcaatc gcgtgttctg tgacacgatg aacagctgta ctttgccatc agaggttaat 1140
ctgtgcaata ccgacatctt caaccccaaa tacgactgta agatcatgac cagcaagact 1200
gatgtcagct cctccgttat aacatcactc ggcgctatcg tgtcttgcta tggcaagacc 1260
aagtgtacag cgtccaataa gaatcggggc attatcaaga cattctccaa cggatgtgac 1320
tacgtgagca acaaaggagt ggacaccgtg tcagtcggaa atacactgta ttacgtgaat 1380
aagcaggagg gcaaatctct ttacgtgaag ggcgaaccaa tcatcaactt ctatgatccc 1440
ctcgtctttc cttctgatga gtttgacgcc tctatttctc aggttaacga gaagatcaat 1500
cagtctctgg cctttatacg caaaagcgat gaactcctgg gatcaggcta cattcccgaa 1560
gctccaaggg acggacaagc gtatgtgcgg aaggatggag agtgggtcct tctgtcaact 1620
ttcctgggcg ggggtgggag cggaggcgga ggttcagggg gtggagggtc aggcggagga 1680
ggcagcgctt ccagggagtt agtggtctcc tacgtcaacg tgaacatggg ccttaagatc 1740
cgtcagttgc tgtggttcca catttccgcc ctaacctttg gcagagagac tgtgctggag 1800
tacctggtgt catttggcgt gtggattcgc actcctccag cagcaagacc cccaaatgcg 1860
cctatcctgt ccacgttacc agagaccacc gttgttgggg gaggtggttc tggagggggc 1920
ggatctggcg gaggtggcag tgggggcggt ggaagtggcg ctagcgtcga gttgctcagc 1980
ttccttccct cggacttctt tccgtccatc agggatctgc tggacacagc cagtgctctc 2040
tatcgggaag cactggaatc tcccgaacac agctctcctc accatacagc ccttcgacaa 2100
gccatactct gctggggcga actgatgaat ctcgccacat gggtagggtc aggggggagc 2160
gactacaagg acgatgacga caaa 2184
<210> 74
<211> 1722
<212> ДНК
<213> Искусственная последовательность
<220>
<223> RSV_F
<400> 74
atggagttgc taatcctcaa agcaaatgca attaccacaa tcctcactgc agtcacattt 60
tgttttgctt ctggtcaaaa catcactgaa gaattttatc aatcaacatg cagtgcagtt 120
agcaaaggct atcttagtgc tctgagaact ggttggtata ccagtgttat aactatagaa 180
ttaagtaata tcaaggaaaa taagtgtaat ggaacagatg ctaaggtaaa attgataaaa 240
caagaattag ataaatataa aaatgctgta acagaattgc agttgctcat gcaaagcaca 300
ccaccaacaa acaatcgagc cagaagagaa ctaccaaggt ttatgaatta tacactcaac 360
aatgccaaaa aaaccaatgt aacattaagc aagaaaagga aaagaagatt tcttggtttt 420
ttgttaggtg ttggatctgc aatcgccagt ggcgttgctg tatctaaggt cctgcaccta 480
gaaggggaag tgaacaagat caaaagtgct ctactatcca caaacaaggc tgtagtcagc 540
ttatcaaatg gagttagtgt cttaaccagc aaagtgttag acctcaaaaa ctatatagat 600
aaacaattgt tacctattgt gaacaagcaa agctgcagca tatcaaatat agaaactgtg 660
atagagttcc aacaaaagaa caacagacta ctagagatta ccagggaatt tagtgttaat 720
gcaggtgtaa ctacacctgt aagcacttac atgttaacta atagtgaatt attgtcatta 780
atcaatgata tgcctataac aaatgatcag aaaaagttaa tgtccaacaa tgttcaaata 840
gttagacagc aaagttactc tatcatgtcc ataataaaag aggaagtctt agcatatgta 900
gtacaattac cactatatgg tgttatagat acaccctgtt ggaaactaca cacatcccct 960
ctatgtacaa ccaacacaaa agaagggtcc aacatctgtt taacaagaac tgacagagga 1020
tggtactgtg acaatgcagg atcagtatct ttcttcccac aagctgaaac atgtaaagtt 1080
caatcaaatc gagtattttg tgacacaatg aacagtttaa cattaccaag tgaaataaat 1140
ctctgcaatg ttgacatatt caaccccaaa tatgattgta aaattatgac ttcaaaaaca 1200
gatgtaagca gctccgttat cacatctcta ggagccattg tgtcatgcta tggcaaaact 1260
aaatgtacag catccaataa aaatcgtgga atcataaaga cattttctaa cgggtgcgat 1320
tatgtatcaa ataaagggat ggacactgtg tctgtaggta acacattata ttatgtaaat 1380
aagcaagaag gtaaaagtct ctatgtaaaa ggtgaaccaa taataaattt ctatgaccca 1440
ttagtattcc cctctgatga atttgatgca tcaatatctc aagtcaacga gaagattaac 1500
cagagcctag catttattcg taaatccgat gaattattac ataatgtaaa tgctggtaaa 1560
tccaccacaa atatcatgat aactactata attatagtga ttatagtaat attgttatca 1620
ttaattgctg ttggactgct cttatactgt aaggccagaa gcacaccagt cacactaagc 1680
aaagatcaac tgagtggtat aaataatatt gcatttagta ac 1722
<210> 75
<211> 20
<212> PRT
<213> Искусственная последовательность
<220>
<223> линкер GS
<400> 75
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| МУТАНТЫ БЕЛКА F RSV | 2016 |
|
RU2788403C2 |
| МУТАНТЫ БЕЛКА F RSV | 2016 |
|
RU2723039C2 |
| АНТИГЕННЫЕ ПОЛИПЕПТИДЫ НА ОСНОВЕ ПОСЛЕДОВАТЕЛЬНОСТИ РЕСПИРАТОРНО-СИНЦИТИАЛЬНОГО ВИРУСА | 2019 |
|
RU2807992C2 |
| БЕЛКИ RSV В ПРЕДШЕСТВУЮЩЕЙ СЛИЯНИЮ КОНФОРМАЦИИ И ИХ ПРИМЕНЕНИЕ | 2014 |
|
RU2761631C2 |
| СКОНСТРУИРОВАННЫЕ МУЛЬТИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА И ДРУГИЕ МУЛЬТИМЕРНЫЕ БЕЛКИ С АСИММЕТРИЧНЫМИ МУТАЦИЯМИ В ОБЛАСТИ CH2-CH3 | 2018 |
|
RU2804031C2 |
| СТАБИЛИЗИРОВАННЫЕ БЕЛКИ F RSV И ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2795459C2 |
| T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, РАСПОЗНАЮЩИЕ МУТАЦИЮ R175H ИЛИ Y220C В P53 | 2020 |
|
RU2830061C2 |
| АНТАГОНИСТЫ NGF ДЛЯ МЕДИЦИНСКОГО ИСПОЛЬЗОВАНИЯ | 2020 |
|
RU2829812C2 |
| СУБЪЕДИНИЧНАЯ ВАКЦИНА ДЛЯ ЛЕЧЕНИЯ ИЛИ ПРЕДОТВРАЩЕНИЯ ИНФЕКЦИИ ДЫХАТЕЛЬНЫХ ПУТЕЙ | 2020 |
|
RU2811991C2 |
| АНТИТЕЛА К РЕЦЕПТОРУ IL4 ДЛЯ ПРИМЕНЕНИЯ В ВЕТЕРИНАРИИ | 2019 |
|
RU2837141C2 |
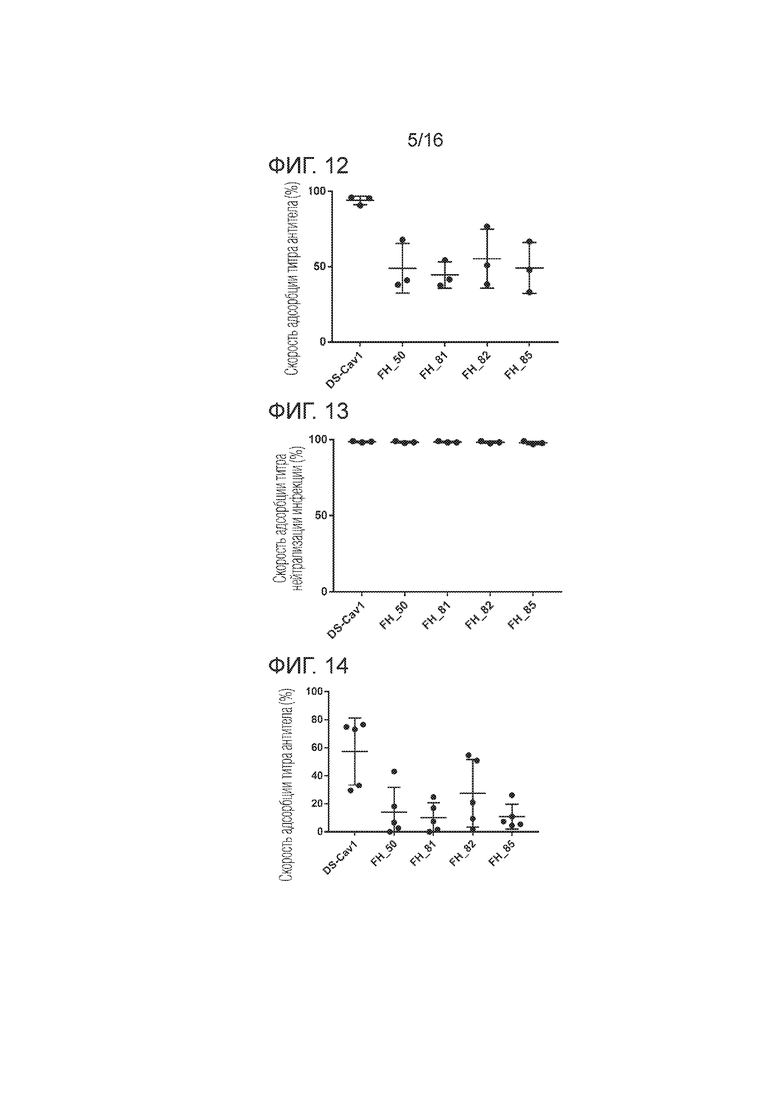
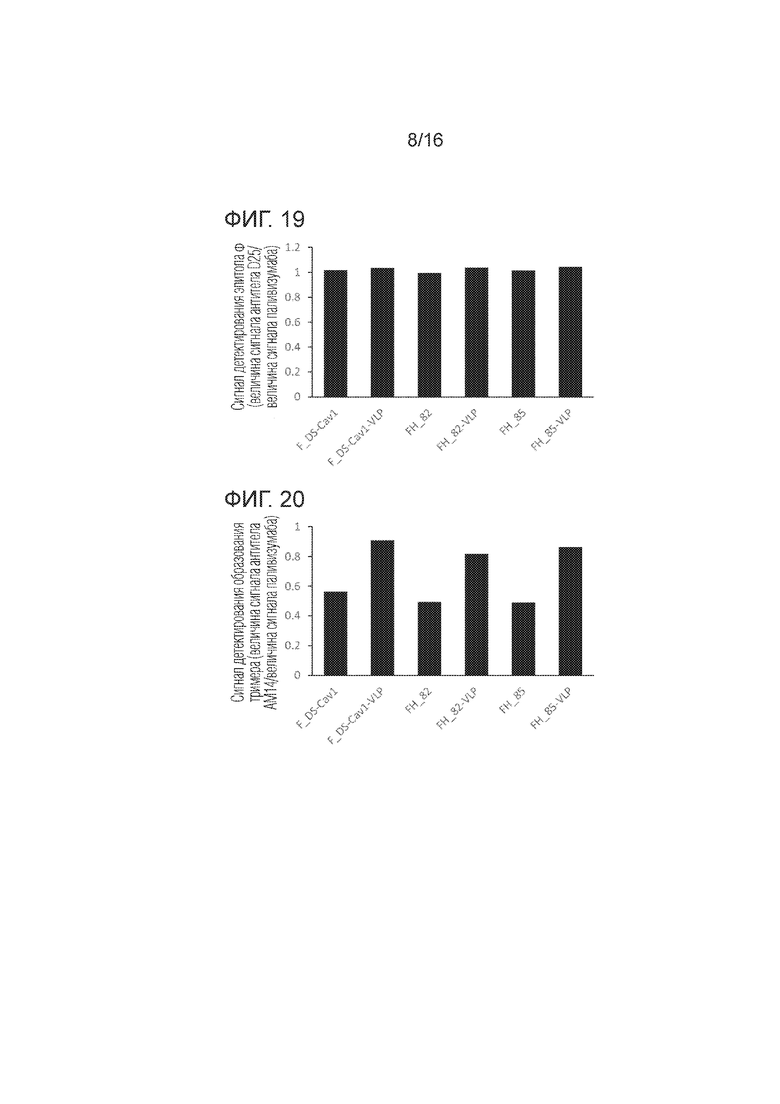


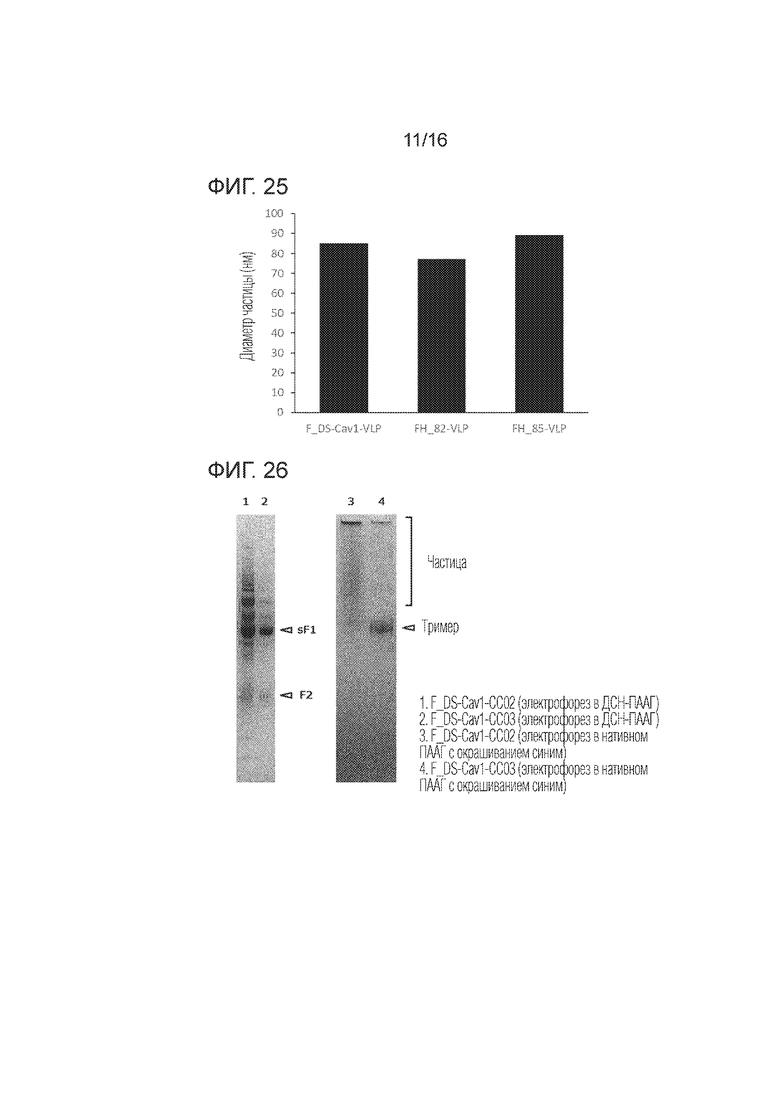
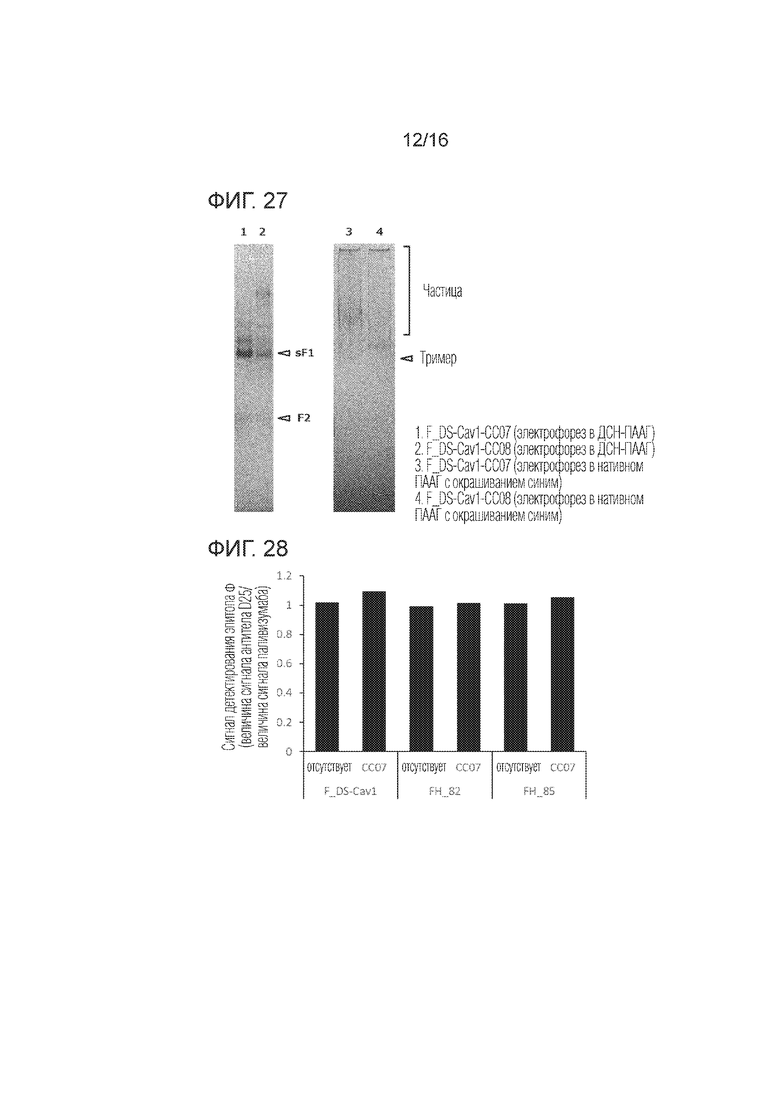
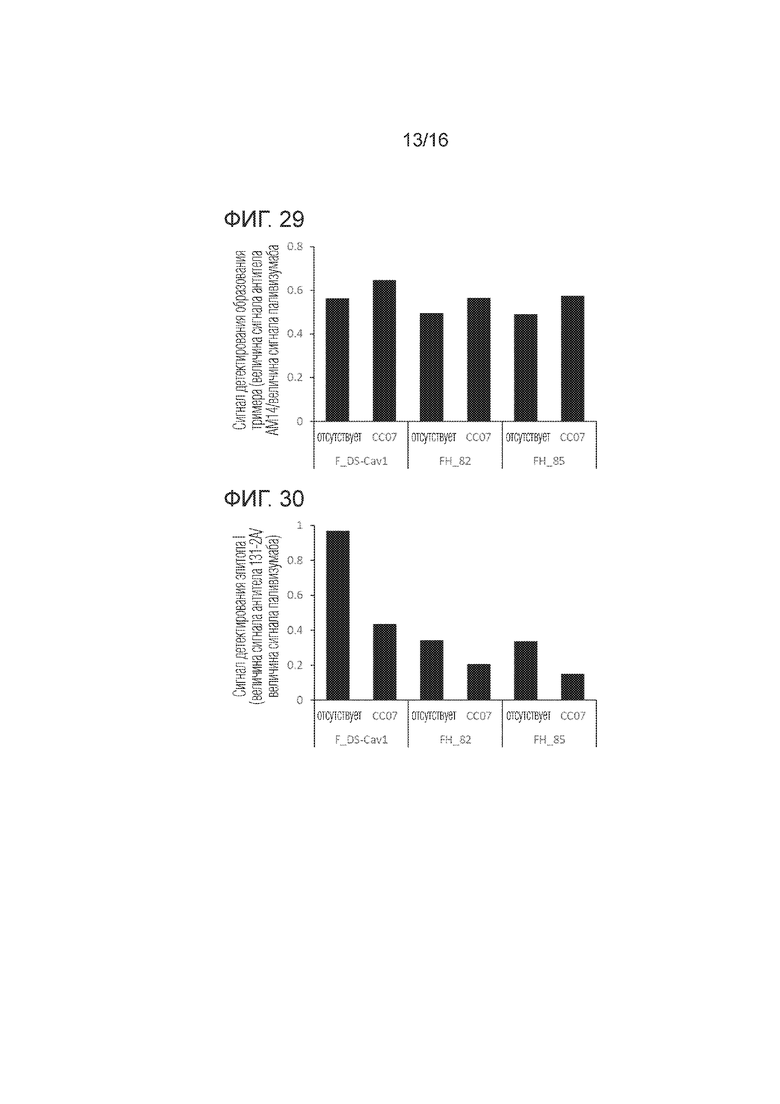
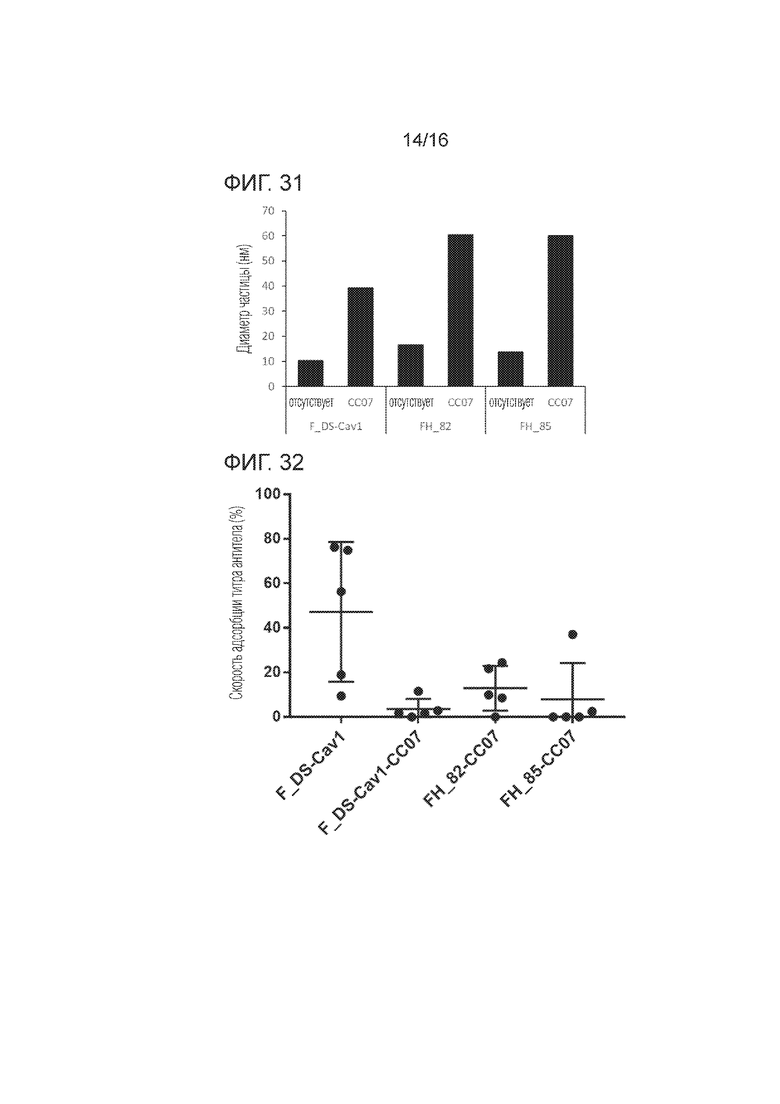
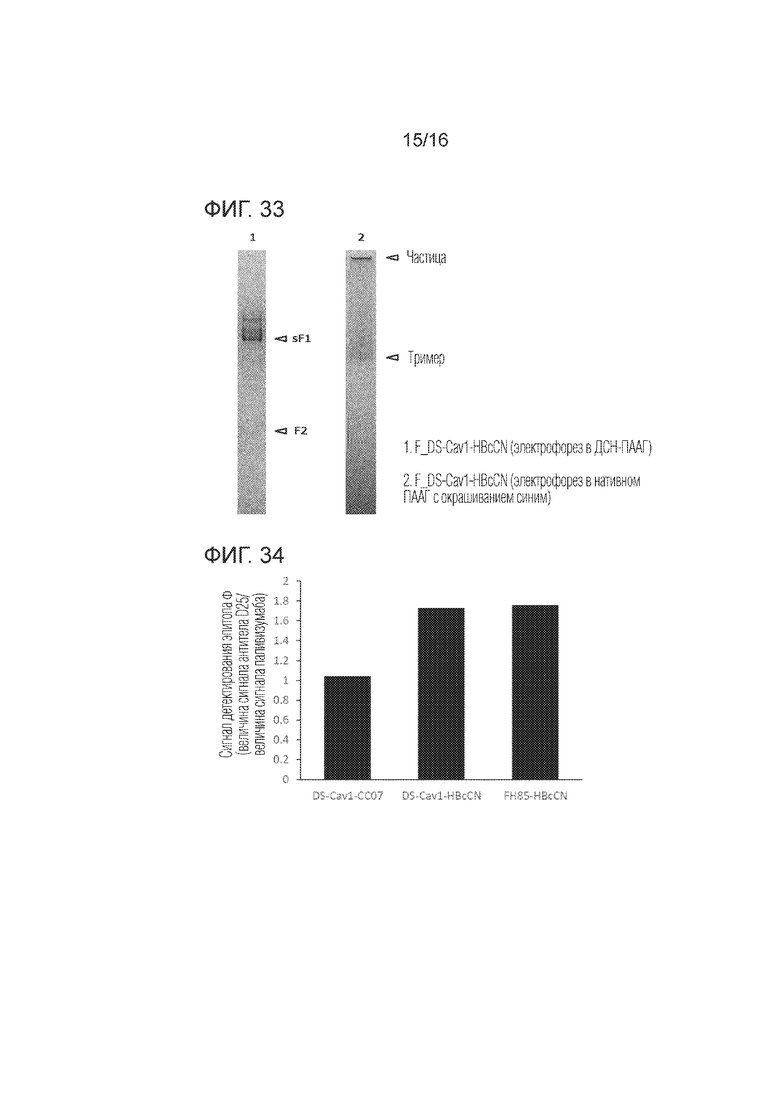
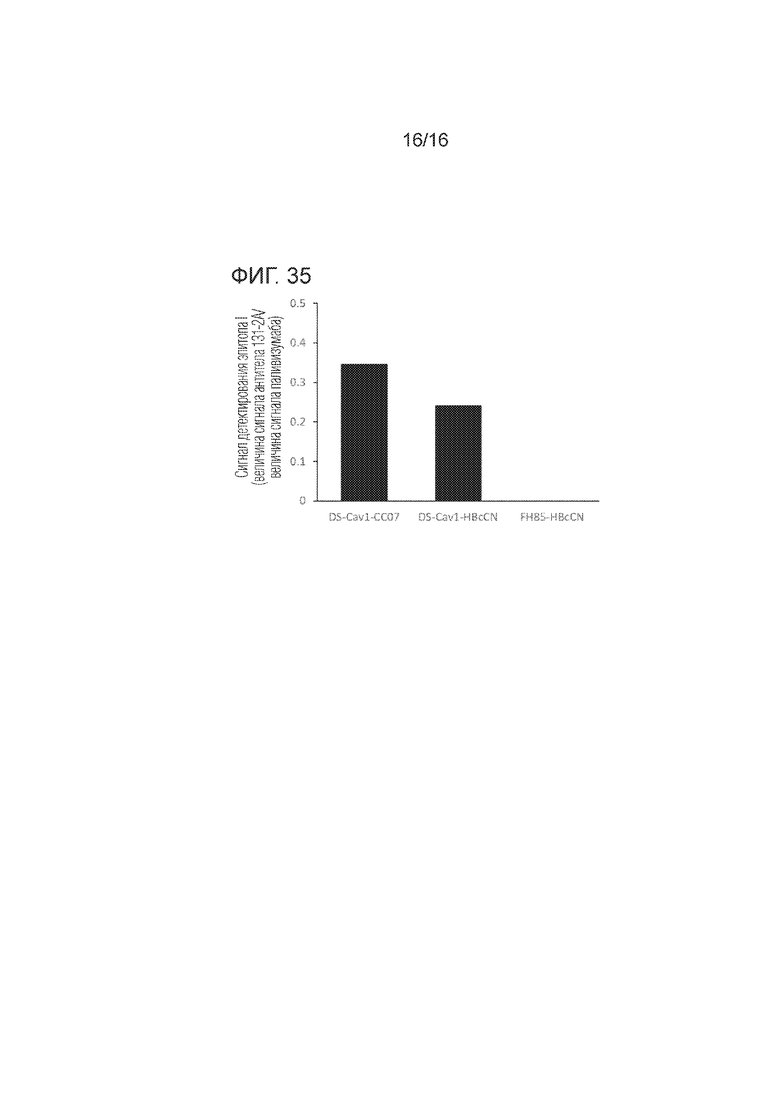
Изобретение относится к биотехнологии и медицине и представляет собой мутантный белок F респираторно-синцитиального вируса (RSV), имеющий мутацию, где мутация представляет собой замену лейцина, соответствующего лейцину в положении 141 аминокислотной последовательности SEQ ID NO: 1, или лейцина, соответствующего лейцину в положении 142, на цистеин, и замену лейцина, соответствующего лейцину в положении 373, на цистеин, с последующим образованием дисульфидной связи между цистеинами. Изобретение касается также слитого белка, мультимера мутантного белка, иммуногенного продукта в виде частиц или иммуногена, фармацевтической композиции и вакцины, содержащих указанный мутантный белок белок F респираторно-синцитиального вируса. Изобретение позволяет эффективно лечить вирусную инфекцию RS. 15 н. и 24 з.п. ф-лы, 35 ил., 9 табл., 9 пр.
1. Мутантный белок F респираторно-синцитиального вируса (RSV), имеющий мутацию, где мутация представляет собой замену лейцина, соответствующего лейцину в положении 141 аминокислотной последовательности SEQ ID NO: 1, или лейцина, соответствующего лейцину в положении 142, на цистеин, и замену лейцина, соответствующего лейцину в положении 373, на цистеин, с последующим образованием дисульфидной связи между цистеинами.
2. Мутантный белок F RSV по п. 1, где мутантный белок F RSV происходит от RSV подтипа A или RSV подтипа B.
3. Мутантный белок F RSV по п. 2, где RSV подтипа А представляет собой штамм RSV A2 или штамм RSV с длинной цепью.
4. Мутантный белок F RSV по п. 2, где RSV подтипа B представляет собой штамм RSV 18537.
5. Мутантный белок F RSV по любому из пп. 1-4, где мутантный белок F RSV содержит аминокислотную последовательность, которая на 85% или более идентична SEQ ID NO: 2, где лейцин, соответствующий лейцину в положении 141 аминокислотной последовательности SEQ ID NO: 1, или лейцин, соответствующий лейцину в положении 142 аминокислотной последовательности SEQ ID NO: 1, и лейцин, соответствующий лейцину в положении 373 аминокислотной последовательности SEQ ID NO: 1, заменены цистеинами с образованием дисульфидной связи между цистеинами, и за счет этого обладающий способностью индуцировать антитело против пре-белка RSV типа F.
6. Мутантный белок F RSV по любому из пп. 1-4, где аминокислота, образующая сайт распознавания фурином, который присутствует на С-концевой стороне области pep27, заменена таким образом, что сайт распознавания фурином не распознается фурином.
7. Мутантный белок F RSV по п. 6, где:
аминокислота, образующая сайт распознавания фурином, заменена неосновной аминокислотой, и аминокислота, образующая сайт распознавания фурином, представляет собой аминокислоту, выбранную из группы, состоящей из аргинина, соответствующего аргинину в положении 133 аминокислотной последовательности SEQ ID NO: 1, аргинина, соответствующего аргинину в положении 135 аминокислотной последовательности SEQ ID NO: 1, и аргинина, соответствующего аргинину в положении 136 аминокислотной последовательности SEQ ID NO: 1.
8. Мутантный белок F RSV по п. 6 или 7, где мутантный белок F RSV, содержит аминокислотную последовательность, где в аминокислотной последовательности, которая на 85% или более идентична SEQ ID NO: 3, где лейцин, соответствующий лейцину в положении 141 аминокислотной последовательности SEQ ID NO: 1, или лейцин, соответствующий лейцину в положении 142 аминокислотной последовательности SEQ ID NO: 1, и лейцин, соответствующий лейцину в положении 373 аминокислотной последовательности SEQ ID NO: 1, заменены цистеинами и, кроме того, аминокислота, выбранная из группы, состоящей из аргинина, соответствующего аргинину в положении 133 аминокислотной последовательности SEQ ID NO: 1, аргинина, соответствующего аргинину в положении 135 аминокислотной последовательности SEQ ID NO: 1, и аргинина, соответствующего аргинину в положении 136 аминокислотной последовательности SEQ ID NO: 1, заменена неосновной аминокислотой, и между цистеинами образуется дисульфидная связь, и за счет этого обладающий способностью индуцировать антитело против пре-белка RSV типа F.
9. Мутантный белок F RSV по п. 7 или 8, где неосновная аминокислота представляет собой аспарагин.
10. Мутантный белок F RSV по любому из пп. 1-9, где треонин, соответствующий треонину в положении 189 аминокислотной последовательности SEQ ID NO: 1, и/или серин, соответствующий серину в положении 190 аминокислотной последовательности SEQ ID NO: 1, заменены гидрофобными аминокислотами.
11. Мутантный белок F RSV по п. 10, где каждая гидрофобная аминокислота независимо выбрана из группы, состоящей из валина, изолейцина и лейцина.
12. Мутантный белок F RSV по любому из пп. 1-11, где, лизин, соответствующий лизину в положении 42 аминокислотной последовательности SEQ ID NO: 1, заменен на аргинин, и/или валин, соответствующий валину в положении 384 аминокислотной последовательности SEQ ID NO: 1, заменен треонином.
13. Мутантный белок F RSV, имеющий мутацию, где мутация представляет собой замену глутаминовой кислоты, соответствующей глутаминовой кислоте в положении 60 аминокислотной последовательности SEQ ID NO: 1, некислотной аминокислотой.
14. Мутантный белок F RSV по п. 13, где некислотная аминокислота выбрана из группы, состоящей из метионина, фенилаланина, лейцина, треонина и серина.
15. Слитый белок F RSV, содержащий:
мутантный белок F RSV по любому из пп. 1-14; и
домен мультимеризации, слитый с С-концом мутантного белка F RSV,
где домен мультимеризации представляет собой домен фолдона.
16. Слитый белок F RSV по п. 15, где домен мультимеризации представляет собой домен фолдона, полученный из фибритина бактериофага Т4.
17. Слитый белок F RSV по п. 15 или п. 16, где домен мультимеризации представляет собой домен фолдона, имеющий аминокислотную последовательность SEQ ID NO: 21.
18. Слитый белок F RSV по любому из пп. 15-17, содержащий аминокислотную последовательность любой из SEQ ID NO: 10-13.
19. Мультимер мутантного белка F RSV, содержащий:
два или более слитых белков по любому из пп. 15-18, связанных посредством домена мультимеризации.
20. Мультимер по п. 19, где мультимер представляет собой тример.
21. Иммуногенный продукт в виде частиц, содержащий:
частицы мутантного белка F RSV по любому из пп. 1-14, где:
мутантный белок F RSV содержит домен, образующий частицы,
два или более мутантных белка F RSV агрегируются посредством домена, образующего частицу, с образованием частицы, указанный образующий частицы домен находится в положении, более близком к эпитопу I, чем к эпитопу Φ на стерической структуре мутантного белка, и указанный образующий частицы домен представляет собой любой из пептида, связывающиегося с каркасными частицами, гидрофобной пептидной цепи и самоассоциирующегося белка.
22. Иммуногенный продукт в виде частиц, содержащий:
частицы мультимера по п. 19 или 20,
где мультимер содержит домен, образующий частицы, два или более мультимера агрегируются посредством домена, образующего частицу, с образованием частицы, и указанный образующий частицы домен находится в положении, более близком к эпитопу I, чем к эпитопу Φ на стерической структуре мультимера, и указанный образующий частицы домен представляет собой любой из пептида, связывающегося с каркасными частицами, гидрофобной пептидной цепи и самоассоциирующегося белка.
23. Иммуногенный продукт в виде частиц по п. 21 или 22, где два или более мутантных белка F RSV или мультимера для получения иммуногенного продукта в виде частиц, в которых домен, образующий частицы, связан с С-концом мутантного белка F RSV или мультмера, агрегируются посредством домена, образующего частицы.
24. Иммуногенный продукт в виде частиц по любому из пп. 21-23, где мультимер представляет собой тример, состоящий из слитого белка F RSV по п. 16.
25. Иммуногенный продукт в виде частиц по п. 24, где образующий частицы домен представляет собой домен Fc, состоящий из аминокислотной последовательности SEQ ID NO: 30, и где домен, образующий частицы, агрегируется посредством связывания с доменом Z белка А, иммобилизованного на VLP, полученной из модифицированного антигена HBs.
26. Иммуногенный продукт в виде частиц по п. 24, где образующий частицы домен представляет собой пептид, состоящий из аминокислотной последовательности SEQ ID NO: 42.
27. Применение слитого белка F RSV для получения иммуногенного продукта в виде частиц, содержащего: мутантный белок F RSV по любому из пп. 1-14 или слитый белок F RSV по любому из пп. 15-18, и образующий частицы домен, слитый с его С-концом, где указанный образующий частицы домен представляет собой любой из пептида, связывающегося с каркасными частицами, гидрофобной пептидной цепи и самоассоциирующегося белка.
28. Применение слитого белка F RSV для получения иммуногенного продукта в виде частиц по п. 27, где домен, образующий частицы, представляет собой домен Fc, состоящий из аминокислотной последовательности SEQ ID NO: 30.
29. Применение слитого белка F RSV для получения иммуногенного продукта в виде частиц по п. 27, где образующий частицы домен представляет собой пептид, состоящий из аминокислотной последовательности SEQ ID NO: 42.
30. Полинуклеотид, кодирующий мутантный белок F RSV по любому из пп. 1-14.
31. Полинуклеотид, кодирующий слитый белок F RSV по любому из пп. 15-18.
32. Экспрессионный кластер, содержащий полинуклеотид по п. 30.
33. Экспрессионный кластер, содержащий полинуклеотид по п. 31.
34. Клетка-хозяин, содержащая экспрессионный кластер по п. 32, где клетка-хозяин экспрессирует мутантные белки F RSV, кодируемые полинуклеотидом из экспрессионного кластера.
35. Клетка-хозяин, содержащая экспрессионный кластер по п. 33, где клетка-хозяин экспрессирует слитые белки F RSV, кодируемые полинуклеотидом из экспрессионного кластера.
36. Иммуноген, содержащий: мутантный белок F RSV по любому из пп. 1-14, слитый белок F RSV по любому из пп. 15-18, мультимер по любому из пп. 19-20 или иммуногенный продукт в виде частиц по любому из пп. 21-26.
37. Фармацевтическая композиция для профилактики или лечения вирусной инфекции RS, содержащая экспрессионный кластер по п. 32 или 33 или иммуноген по п. 36.
38. Вакцина RSV, содержащая экспрессионный кластер по п. 32 или 33 или иммуноген по п. 36.
39. Фармацевтическая композиция по п. 37, которая предназначена для введения человеку.
40. Вакцина RSV по п.38, которая предназначена для введения человеку.
| WO 2014160463 A1, 02.10.2014 | |||
| WO 2017109629 A1, 29.06.2017 | |||
| WO 2017172890 A1, 05.10.2017 | |||
| КОМПОЗИЦИИ БЕЛКА F RSV И СПОСОБЫ ИХ ПОЛУЧЕНИЯ | 2010 |
|
RU2585227C2 |
Авторы
Даты
2023-11-21—Публикация
2020-12-23—Подача