ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННЫЕ ЗАЯВКИ
[0001] Данная заявка заявляет приоритет относительно заявки США №62/820101, поданной 18 марта 2019 г., которая включена в данный документ посредством ссылки в полном ее объеме и для всех целей.
ССЫЛКА НА ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ, ПРЕДСТАВЛЕННЫЙ В ТЕКСТОВОМ ФАЙЛЕ ПОСРЕДСТВОМ EPS WEB
[0002] Перечень последовательностей записан в файле 544640SEQLIST.txt, имеющем размер 75,7 килобайт, был создан 16 марта 2020 г. и включен в данный документ посредством ссылки.
УРОВЕНЬ ТЕХНИКИ
[0003] Аномальная агрегация или фибриллизация белков является определяющим признаком многих заболеваний, в частности, ряда нейродегенеративных заболеваний, таких как болезнь Альцгеймера (БА), болезнь Паркинсона (БП), лобно-височная деменция (ЛВД), боковой амиотрофический склероз (БАС), хроническая травматическая энцефалопатия (ХТЭ), болезнь Крейтцфельда - Якоба (БКЯ) и другие. Во многих из данных заболеваний фибриллизация определенных белков в нерастворимые агрегаты является не только характерным признаком заболевания, но также считается причинным фактором нейротоксичности. Кроме того, данные заболевания характеризуются распространением совокупной патологии через центральную нервную систему в соответствии со стереотипными паттернами - процессом, который коррелирует с прогрессированием заболевания. Таким образом, идентификация генов и генетических путей, которые модифицируют процессы аномальной агрегации белков или распространения агрегатов от клетки к клетке, имеет большое значение для лучшего понимания этиологии нейродегенеративных заболеваний, а также для разработки стратегий терапевтического вмешательства.
КРАТКОЕ ОПИСАНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
[0004] В данном документе представлены способы скрининга в отношении генетических уязвимостей, связанных с агрегацией тау-белка. В данном документе также представлены биосенсорные клетки Cas-тау или популяции таких клеток, а также in vitro культуры биосенсорных клеток Cas-тау и культуральная среда. В данном документе также представлены биосенсорные клетки CRISPR/Cas медиатора синергической активации (SAM)-тау или популяции таких клеток, а также in vitro культуры биосенсорных клеток SAM-тау и культуральная среда.
[0005] В одном аспекте представлены способы скрининга в отношении генетических уязвимостей, связанных с агрегацией тау-белка. Некоторые из таких способов включают в себя: (а) обеспечение положительной в отношении агрегации популяции клеток и обеспечение отрицательной в отношении агрегации популяции клеток, при этом каждая популяция клеток содержит белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, при этом в указанной положительной в отношении агрегации популяции клеток указанный первый содержащий повторы домен тау-белка, связанный с первым репортером, и указанный второй содержащий повторы домен тау-белка, связанный со вторым репортером, присутствуют стабильно в агрегированном состоянии, и при этом в указанной отрицательной в отношении агрегации популяции клеток указанный первый содержащий повторы домен тау-белка, связанный с первым репортером, и указанный второй содержащий повторы домен тау-белка, связанный со вторым репортером, не присутствуют стабильно в агрегированном состоянии; (b) введение в каждую популяцию клеток библиотеки, содержащей множество уникальных гидовых РНК, которые нацелены на множество генов, при этом указанное множество уникальных гидовых РНК образует комплексы с указанным белком Cas, и указанный белок Cas расщепляет указанное множество генов, что приводит к нокауту генной функции; и (с) определение содержания каждой из указанного множества уникальных гидовых РНК во множестве моментов времени в исследовании с течением времени в каждой популяции клеток, при этом истощение гидовой РНК в указанной положительной в отношении агрегации популяции клеток, но не в указанной отрицательной в отношении агрегации популяции клеток, или паттерн более резкого истощения гидовой РНК в исследовании с течением времени в указанной положительной в отношении агрегации популяции клеток по сравнению с указанной отрицательной в отношении агрегации популяцией клеток указывает на то, что ген, на который нацелена указанная гидовая РНК, проявляет синтетическую летальность с агрегатами тау-белка и является генетической уязвимостью, связанной с агрегацией тау-белка, или является потенциальной генетической уязвимостью, связанной с агрегацией тау-белка (например, кандидатом для дальнейшего тестирования с помощью вторичных скринингов). Гидовые РНК, нацеленные на такие гены, вызывают избирательную гибель клеток в популяции клеток Аг[+], но не в популяции клеток Аг[-].
[0006] В некоторых из таких способов первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка представляют собой содержащий повторы домен тау-белка человека. В некоторых из таких способов первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат мутацию, способствующую агрегации. Необязательно, первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат мутацию тау-белка P301S. В некоторых из таких способов первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат домен тау-белка с четырьмя повторами. В некоторых из таких способов первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат последовательность с SEQ ID NO: 11. В некоторых из таких способов первый содержащий повторы домен тау-белка и второй содержащий повторы домен тау-белка являются одинаковыми. В некоторых из таких способов первый содержащий повторы домен тау-белка и второй содержащий повторы домен тау-белка являются одинаковыми, и каждый из них содержит домен тау-белка с четырьмя повторами, содержащий мутацию тау-белка P301S.
[0007] В некоторых из таких способов первый репортер и второй репортер представляют собой флуоресцентные белки. Необязательно, первый репортер и второй репортер представляют собой пару с резонансным переносом энергии флуоресценции (FRET). Необязательно, первый репортер представляет собой голубой флуоресцентный белок (CFP), а второй репортер представляет собой желтый флуоресцентный белок (YFP).
[0008] В некоторых из таких способов белок Cas представляет собой белок Cas9. Необязательно, белок Cas представляет собой Cas9 из Streptococcus pyogenes. Необязательно, белок Cas содержат последовательность с SEQ ID NO: 21. Необязательно, белок Cas кодируется кодирующей последовательностью, содержащей последовательность, указанную в SEQ ID NO: 22.
[0009] В некоторых из таких способов белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, стабильно экспрессируются в указанных популяциях клеток. В некоторых из таких способов нуклеиновые кислоты, кодирующие белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, геномно интегрированы в указанные популяции клеток.
[0010] В некоторых из таких способов клетки представляют собой эукариотические клетки. Необязательно, клетки представляют собой клетки млекопитающих. Необязательно, клетки представляют собой клетки человека. Необязательно, клетки представляют собой клетки НЕК293Т.
[0011] В некоторых из таких способов множество уникальных гидовых РНК вводят в концентрации, выбранной таким образом, чтобы большинство клеток получали только по одной из уникальных гидовых РНК. В некоторых из таких способов множество уникальных гидовых РНК нацелено на 100 или большее число генов, 1000 или большее число генов, или 10000 или большее число генов. В некоторых из таких способов указанная библиотека представляет собой полногеномную библиотеку. В некоторых способах нацеливание происходит в среднем на множество целевых последовательностей в каждом из целевого множества генов. Необязательно, нацеливание происходит в среднем на по меньшей мере три целевые последовательности в каждом из целевого множества генов. Необязательно, нацеливание происходит в среднем на от около трех до около шести целевых последовательностей (например, на около трех, на около четырех или на около шести) в каждом из целевого множества генов.
[0012] В некоторых из таких способов каждая гидовая РНК нацелена на конститутивный экзон. Необязательно, каждая гидовая РНК нацелена на конститутивный 5'-экзон. В некоторых из таких способов каждая гидовая РНК нацелена на первый экзон, на второй экзон или на третий экзон.
[0013] В некоторых из таких способов множество уникальных гидовых РНК вводят в указанные популяции клеток с помощью вирусной трансдукции. Необязательно, каждое из множества уникальных гидовых РНК находится в отдельном вирусном векторе. В некоторых из таких способов множество уникальных гидовых РНК вводят в указанные популяции клеток с помощью лентивирусной трансдукции. В некоторых из таких способов указанные популяции клеток инфицированы с показателем множественности заражения, составляющим меньше чем около 0,3.
[0014] В некоторых из таких способов множество уникальных гидовых РНК вводят в указанные популяции клеток вместе с маркером отбора, и стадия (b) дополнительно включает в себя отбор клеток, которые содержат маркер отбора. Необязательно, маркер отбора придает устойчивость к лекарственному средству. Необязательно, маркер отбора придает устойчивость к пуромицину или зеоцину. Необязательно, маркер отбора выбран из неомицинфосфотрансферазы, гигромицин-В-фосфотрансферазы, пуромицин-N-ацетилтрансферазы и бластицидин-S-дезаминазы. Необязательно, маркер отбора выбран из неомицинфосфотрансферазы, гигромицин-В-фосфотрансферазы, пуромицин-N-ацетилтрансферазы, бластицидин-S-дезаминазы и белка устойчивости к блеомицину.
[0015] В некоторых из таких способов каждая из указанных популяций клеток, в которые на стадии (b) вводят множество уникальных гидовых РНК, включает в себя больше чем около 500 клеток на уникальную гидовую РНК.
[0016] В некоторых из таких способов указанное исследование с течением времени на стадии (с) имеет продолжительность больше чем около 1-й недели. Необязательно, указанное исследование с течением времени на стадии (с) имеет продолжительность больше чем около 2-х недель. В некоторых из таких способов указанное исследование с течением времени на стадии (с) включает в себя от около 10 до около 15 удвоений клеток. В некоторых из таких способов указанное множество моментов времени на стадии (с) включает в себя по меньшей мере три момента времени. Необязательно, указанное множество моментов времени на стадии (с) включает в себя около четырех моментов времени или около шести моментов времени. В некоторых из таких способов между каждым моментом времени на стадии (с) проходит более 1-х суток. Необязательно, между каждым моментом времени на стадии (с) проходит более 2-х суток. Необязательно, между каждым моментом времени на стадии (с) проходит от около 3-х суток до около 4 суток.
[0017] В некоторых из таких способов считается, что ген проявляет синтетическую летальность с агрегатами тау-белка (или ожидается, что ген проявляет синтетическую летальность с агрегатами тау-белка) на стадии (с), если гидовая РНК, нацеленная на данный ген, истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток. Необязательно, считается, что ген проявляет синтетическую летальность с агрегатами тау-белка на стадии (с) (или ожидается, что ген проявляет синтетическую летальность с агрегатами тау-белка), если гидовая РНК, нацеленная на данный ген, имеет паттерн более резкого истощения с течением времени в положительной в отношении агрегации популяции клеток по сравнению с отрицательной в отношении агрегации популяцией клеток.
[0018] В некоторых из таких способов гидовая РНК считается истощенной на стадии (с), если содержание данной гидовой РНК в каждый из моментов времени является меньшим или равным содержанию данной гидовой РНК в предшествующий момент времени. В некоторых из таких способов гидовая РНК считается истощенной на стадии (с), если содержание данной гидовой РНК в каждый из моментов времени после второго момента времени является меньшим или равным содержанию в момент времени, на два момента времени предшествующий данному моменту времени.
[0019] В некоторых из таких способов считается, что ген проявляет синтетическую летальность с агрегатами тау-белка на стадии (с) (или ожидается, что ген проявляет синтетическую летальность с агрегатами тау-белка), если больше чем 30% гидовых РНК в данной библиотеке, нацеленных на данный ген, истощено в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток. Необязательно, считается, что ген проявляет синтетическую летальность с агрегатами тау-белка на стадии (с) (или ожидается, что ген будет проявлять синтетическую летальность с агрегатами тау-белка) в любой из следующих ситуаций: (1) в библиотеке есть одна гидовая РНК, которая нацелена на данный ген, и данная одна гидовая РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (2) в библиотеке есть две гидовые РНК, которые нацелены на данный ген, и по меньшей мере одна из данных двух гидовых РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (3) в библиотеке есть три гидовые РНК, которые нацелены на данный ген, и по меньшей мере одна из данных трех гидовых РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (4) в библиотеке есть четыре гидовые РНК, которые нацелены на данный ген, и по меньшей мере две из данных четырех гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (5) в библиотеке есть пять гидовых РНК, которые нацелены на данный ген, и по меньшей мере две из данных пяти гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; и (6) в библиотеке есть шесть гидовых РНК, которые нацелены на данный ген, и по меньшей мере три из данных шести гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток.
[0020] В некоторых из таких способов указанный способ повторяется по меньшей мере три раза в по меньшей мере трех различных экспериментах, и считается, что ген проявляет синтетическую летальность с агрегатами тау-белка (или ожидается, что ген проявляет синтетическую летальность с агрегатами тау-белка), если данный ген, как считается, проявляет синтетическую летальность с агрегатами тау-белка в больше чем около 50% из указанных по меньшей мере трех различных экспериментов.
[0021] В некоторых из таких способов указанное исследование с течением времени на стадии (с) имеет продолжительность больше чем около 2-х недель, при этом указанное множество моментов времени на стадии (с) включает в себя около шести временных точек, при этом между каждым моментом времени на стадии (с) проходит от около 3-х суток до около 4-х суток, при этом гидовая РНК считается истощенной на стадии (с), если содержание данной гидовой РНК в каждый момент времени после второго момента времени является меньшим или равным содержанию в момент времени, на два момента времени предшествующий данному моменту времени, и при этом считается, что ген проявляет синтетическую летальность с агрегатами тау-белка на стадии (с) (или ожидается, что ген проявляет синтетическую летальность с агрегатами тау-белка), если больше чем 30% гидовых РНК в данной библиотеке, нацеленных на данный ген, истощено в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток.
[0022] В других из таких способов используются библиотеки гидовых РНК активации CRISPR (медиатор синергической активации CRISPR/Cas (SAM)). Некоторые из таких способов включают в себя: (а) обеспечение положительной в отношении агрегации популяции клеток и обеспечение отрицательной в отношении агрегации популяции клеток, при этом каждая популяция клеток содержит химерный белок Cas, содержащий белок Cas с инактивированной нуклеазной активностью (т.е. каталитически неактивный белок Cas), слитый с одним или большим числом доменов активации транскрипции, химерный адаптерный белок, содержащий адаптерный белок, слитый с одним или большим числом доменов активации транскрипции, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, при этом в указанной положительной в отношении агрегации популяции клеток указанный первый содержащий повторы домен тау-белка, связанный с первым репортером, и указанный второй содержащий повторы домен тау-белка, связанный со вторым репортером, присутствуют стабильно в агрегированном состоянии, и при этом в указанной отрицательной в отношении агрегации популяции клеток указанный первый содержащий повторы домен тау-белка, связанный с первым репортером, и указанный второй содержащий повторы домен тау-белка, связанный со вторым репортером, не присутствуют стабильно в агрегированном состоянии; (b) введение в каждую популяцию клеток библиотеки, содержащей множество уникальных гидовых РНК, которые нацелены на множество генов, при этом указанное множество уникальных гидовых РНК образует комплексы с указанным химерным белком Cas и указанным химерным адаптерный белком, и указанные комплексы активируют транскрипцию указанного множества генов, что приводит к повышенной генной экспрессии; и (с) определение содержания каждой из указанного множества уникальных гидовых РНК во множестве моментов времени в исследовании с течением времени в каждой популяции клеток, при этом истощение гидовой РНК в указанной положительной в отношении агрегации популяции клеток, но не в указанной отрицательной в отношении агрегации популяции клеток, или паттерн более резкого истощения гидовой РНК в исследовании с течением времени в указанной положительной в отношении агрегации популяции клеток по сравнению с указанной отрицательной в отношении агрегации популяцией клеток указывает на то, что активация гена, на который нацелена указанная гидовая РНК, проявляет синтетическую летальность с агрегатами тау-белка и является генетической уязвимостью, связанной с агрегацией тау-белка, или является потенциальной генетической уязвимостью, связанной с агрегацией тау-белка (например, кандидатом для дальнейшего тестирования с помощью вторичных скринингов).
[0023] В некоторых из таких способов первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка представляют собой содержащий повторы домен тау-белка человека. В некоторых из таких способов первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат мутацию, способствующую агрегации. Необязательно, первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат мутацию тау-белка P301S. В некоторых из таких способов первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат домен тау-белка с четырьмя повторами. В некоторых из таких способов первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат последовательность с SEQ ID NO: 11. В некоторых из таких способов первый содержащий повторы домен тау-белка и второй содержащий повторы домен тау-белка являются одинаковыми. В некоторых из таких способов первый содержащий повторы домен тау-белка и второй содержащий повторы домен тау-белка являются одинаковыми, и каждый из них содержит домен тау-белка с четырьмя повторами, содержащий мутацию тау-белка P301S.
[0024] В некоторых из таких способов первый репортер и второй репортер представляют собой флуоресцентные белки. Необязательно, первый репортер и второй репортер представляют собой пару с резонансным переносом энергии флуоресценции (FRET). Необязательно, первый репортер представляет собой голубой флуоресцентный белок (CFP), а второй репортер представляет собой желтый флуоресцентный белок (YFP).
[0025] В некоторых из таких способов белок Cas представляет собой белок Cas9. Необязательно, белок Cas представляет собой Cas9 из Streptococcus pyogenes. В некоторых из таких способов химерный белок Cas содержит белок Cas с инактивированной нуклеазной активностью, слитый с доменом активации транскрипции VP64, необязательно при этом указанный химерный белок Cas содержит, в направлении от N-конца до С-конца: белок Cas с инактивированной нуклеазной активностью; сигнал ядерной локализации; и домен активатора транскрипции VP64. В некоторых из таких способов адаптерный белок представляет собой белок оболочки MS2, а один или большее число доменов активации транскрипции в указанном химерном адаптерном белке содержат домен активации транскрипции р65 и домен активации транскрипции HSF1, необязательно при этом указанный химерный адаптерный белок содержит, в направлении от N-конца до С-конца: белок оболочки MS2; сигнал ядерной локализации; домен активации транскрипции р65; и домен активации транскрипции HSF1. В некоторых из таких способов химерный белок Cas содержит последовательность с SEQ ID NO: 36, необязательно при этом указанный химерный белок Cas кодируется кодирующей последовательностью, содержащей последовательность, указанную в SEQ ID NO: 38. В некоторых из таких способов химерный адаптерный белок содержит последовательность с SEQ ID NO: 37, необязательно при этом указанный химерный адаптерный белок кодируется кодирующей последовательностью, содержащей последовательность, указанную в SEQ ID NO: 39.
[0026] В некоторых из таких способов химерный белок Cas, химерный адаптерный белок, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, стабильно экспрессируются в популяции клеток. В некоторых из таких способов нуклеиновые кислоты, кодирующие химерный белок Cas, химерный адаптерный белок, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, геномно интегрированы в указанные популяции клеток.
[0027] В некоторых из таких способов клетки представляют собой эукариотические клетки. Необязательно, клетки представляют собой клетки млекопитающих. Необязательно, клетки представляют собой клетки человека. Необязательно, клетки представляют собой клетки НЕК293Т.
[0028] В некоторых из таких способов множество уникальных гидовых РНК вводят в концентрации, выбранной таким образом, чтобы большинство клеток получали только по одной из уникальных гидовых РНК. В некоторых из таких способов множество уникальных гидовых РНК нацелено на 100 или большее число генов, 1000 или большее число генов, или 10000 или большее число генов. В некоторых из таких способов указанная библиотека представляет собой полногеномную библиотеку. В некоторых способах нацеливание происходит в среднем на множество целевых последовательностей в каждом из целевого множества генов. Необязательно, нацеливание происходит в среднем на по меньшей мере три целевые последовательности в каждом из целевого множества генов. Необязательно, нацеливание происходит в среднем на от около трех до около шести целевых последовательностей (например, на около трех, на около четырех или на около шести) в каждом из целевого множества генов. Необязательно, нацеливание происходит в среднем на около три целевые последовательностей в каждом из целевого множества генов.
[0029] В некоторых из таких способов каждая гидовая РНК нацелена на целевую последовательность для гидовой РНК в пределах 200 п. о. в направлении к 5' от сайта начала транскрипции. В некоторых из таких способов каждая гидовая РНК содержит один или большее число адаптер-связывающих элементов, с которыми может специфически связываться химерный адаптерный белок. Необязательно, каждая гидовая РНК содержит два адаптер-связывающих элемента, с которыми может специфически связываться химерный адаптерный белок. Необязательно, первый адаптер-связывающий элемент находится внутри первой петли каждой из одной или большего числа гидовых РНК, а второй адаптер-связывающий элемент находится внутри второй петли каждой из одной или большего числа гидовых РНК. Необязательно, адаптер-связывающий элемент содержит последовательность, указанную в SEQ ID NO: 33. Необязательно, каждая из одной или большего числа гидовых РНК представляет собой единую гидовую РНК, содержащую часть CRISPR-PHK (крРНК), слитую с частью трансактивирующей CRISPR-PHK (тракрРНК), и первая петля представляет собой тетрапетлю, соответствующую остаткам 13-16 из последовательности с SEQ ID NO: 17, 19, 30 или 31, а вторая петля представляет собой шпильку 2, соответствующую остаткам 53-56 из последовательности с SEQ ID NO: 17, 19, 30 или 31.
[0030] В некоторых из таких способов множество уникальных гидовых РНК вводят в указанные популяции клеток с помощью вирусной трансдукции. Необязательно, каждое из множества уникальных гидовых РНК находится в отдельном вирусном векторе. В некоторых из таких способов множество уникальных гидовых РНК вводят в указанные популяции клеток с помощью лентивирусной трансдукции. В некоторых из таких способов указанные популяции клеток инфицированы с показателем множественности заражения, составляющим меньше чем около 0,3.
[0031] В некоторых из таких способов множество уникальных гидовых РНК вводят в указанные популяции клеток вместе с маркером отбора, и стадия (b) дополнительно включает в себя отбор клеток, которые содержат маркер отбора. Необязательно, маркер отбора придает устойчивость к лекарственному средству. Необязательно, маркер отбора придает устойчивость к пуромицину или зеоцину. Необязательно, маркер отбора выбран из неомицинфосфотрансферазы, гигромицин-В-фосфотрансферазы, пуромицин-N-ацетилтрансферазы и бластицидин-S-дезаминазы. Необязательно, маркер отбора выбран из неомицинфосфотрансферазы, гигромицин-В-фосфотрансферазы, пуромицин-N-ацетилтрансферазы, бластицидин-S-дезаминазы и белка устойчивости к блеомицину.
[0032] В некоторых из таких способов каждая из указанных популяций клеток, в которые на стадии (b) вводят множество уникальных гидовых РНК, включает в себя больше чем около 500 клеток на уникальную гидовую РНК.
[0033] В некоторых из таких способов указанное исследование с течением времени на стадии (с) имеет продолжительность больше чем около 1-й недели. Необязательно, указанное исследование с течением времени на стадии (с) имеет продолжительность больше чем около 2-х недель. В некоторых из таких способов указанное исследование с течением времени на стадии (с) включает в себя от около 10 до около 15 удвоений клеток. В некоторых из таких способов указанное множество моментов времени на стадии (с) включает в себя по меньшей мере три момента времени. Необязательно, указанное множество моментов времени на стадии (с) включает в себя около четырех моментов времени или около шести моментов времени. В некоторых из таких способов между каждым моментом времени на стадии (с) проходит более 1-х суток. Необязательно, между каждым моментом времени на стадии (с) проходит более 2-х суток. Необязательно, между каждым моментом времени на стадии (с) проходит от около 3-х суток до около 4 суток.
[0034] В некоторых из таких способов считается, что ген проявляет синтетическую летальность с агрегатами тау-белка на стадии (с) (или ожидается, что ген проявляет синтетическую летальность с агрегатами тау-белка), если гидовая РНК, нацеленная на данный ген, истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток. Необязательно, считается, что ген проявляет синтетическую летальность с агрегатами тау-белка на стадии (с) (или ожидается, что ген проявляет синтетическую летальность с агрегатами тау-белка), если гидовая РНК, нацеленная на данный ген, имеет паттерн более резкого истощения с течением времени в положительной в отношении агрегации популяции клеток по сравнению с отрицательной в отношении агрегации популяцией клеток.
[0035] В некоторых из таких способов гидовая РНК считается истощенной на стадии (с), если содержание данной гидовой РНК в каждый из моментов времени является меньшим или равным содержанию данной гидовой РНК в предшествующий момент времени. В некоторых из таких способов гидовая РНК считается истощенной на стадии (с), если содержание данной гидовой РНК в каждый из моментов времени после второго момента времени является меньшим или равным содержанию в момент времени, на два момента времени предшествующий данному моменту времени.
[0036] В некоторых из таких способов считается, что ген проявляет синтетическую летальность с агрегатами тау-белка на стадии (с) (или ожидается, что ген проявляет синтетическую летальность с агрегатами тау-белка), если больше чем 30% гидовых РНК в данной библиотеке, нацеленных на данный ген, истощено в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток. Необязательно, считается, что ген проявляет синтетическую летальность с агрегатами тау-белка на стадии (с) (или ожидается, что ген будет проявлять синтетическую летальность с агрегатами тау-белка) в любой из следующих ситуаций: (1) в библиотеке есть одна гидовая РНК, которая нацелена на данный ген, и данная одна гидовая РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (2) в библиотеке есть две гидовые РНК, которые нацелены на данный ген, и по меньшей мере одна из данных двух гидовых РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (3) в библиотеке есть три гидовые РНК, которые нацелены на данный ген, и по меньшей мере одна из данных трех гидовых РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (4) в библиотеке есть четыре гидовые РНК, которые нацелены на данный ген, и по меньшей мере две из данных четырех гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (5) в библиотеке есть пять гидовых РНК, которые нацелены на данный ген, и по меньшей мере две из данных пяти гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; и (6) в библиотеке есть шесть гидовых РНК, которые нацелены на данный ген, и по меньшей мере три из данных шести гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток.
[0037] В некоторых из таких способов указанный способ повторяется по меньшей мере три раза в по меньшей мере трех различных экспериментах, и считается, что ген проявляет синтетическую летальность с агрегатами тау-белка (или ожидается, что ген проявляет синтетическую летальность с агрегатами тау-белка), если данный ген, как считается, проявляет синтетическую летальность с агрегатами тау-белка в больше чем около 50% из указанных по меньшей мере трех различных экспериментов.
[0038] В некоторых из таких способов указанное исследование с течением времени на стадии (с) имеет продолжительность больше чем около 2-х недель, при этом указанное множество моментов времени на стадии (с) включает в себя около шести временных точек, при этом между каждым моментом времени на стадии (с) проходит от около 3-х суток до около 4-х суток, при этом гидовая РНК считается истощенной на стадии (с), если содержание данной гидовой РНК в каждый момент времени после второго момента времени является меньшим или равным содержанию в момент времени, на два момента времени предшествующий данному моменту времени, и при этом считается, что ген проявляет синтетическую летальность с агрегатами тау-белка на стадии (с) (или ожидается, что ген проявляет синтетическую летальность с агрегатами тау-белка), если больше чем 30% гидовых РНК в данной библиотеке, нацеленных на данный ген, истощено в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток.
[0039] В другом аспекте представлены биосенсорные клетки Cas-тау и популяция таких клеток, например, популяция из одной или большего числа клеток, содержащих белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером.
[0040] В некоторых из таких клеток первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка представляют собой содержащий повторы домен тау-белка человека. В некоторых из таких клеток первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат мутацию, способствующую агрегации. Необязательно, первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат мутацию тау-белка P301S. В некоторых из таких клеток первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат домен тау-белка с четырьмя повторами. В некоторых из таких клеток первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат последовательность с SEQ ID NO: 11. В некоторых из таких клеток первый содержащий повторы домен тау-белка и второй содержащий повторы домен тау-белка являются одинаковыми. В некоторых из таких клеток первый содержащий повторы домен тау-белка и второй содержащий повторы домен тау-белка являются одинаковыми, и каждый из них содержит домен тау-белка с четырьмя повторами, содержащий мутацию тау-белка P301S.
[0041] В некоторых из таких клеток первый репортер и второй репортер представляют собой флуоресцентные белки. Необязательно, первый репортер и второй репортер представляют собой пару с резонансным переносом энергии флуоресценции (FRET). Необязательно, первый репортер представляет собой голубой флуоресцентный белок (CFP), а второй репортер представляет собой желтый флуоресцентный белок (YFP).
[0042] В некоторых из таких клеток белок Cas представляет собой белок Cas9. Необязательно, белок Cas представляет собой Cas9 из Streptococcus pyogenes. Необязательно, белок Cas содержат последовательность с SEQ ID NO: 21. Необязательно, белок Cas кодируется кодирующей последовательностью, содержащей последовательность, указанную в SEQ ID NO: 22.
[0043] В некоторых из таких клеток белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, стабильно экспрессируются в указанной клетке. В некоторых из таких клеток нуклеиновые кислоты, кодирующие белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, геномно интегрированы в указанную клетку.
[0044] Некоторые из таких клеток представляют собой эукариотические клетки. Необязательно, клетки представляют собой клетки млекопитающих. Необязательно, клетки представляют собой клетки человека. Необязательно, клетки представляют собой клетки НЕК293Т. Необязательно, клетки находятся in vitro.
[0045] В некоторых из таких клеток первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, не присутствуют стабильно в агрегированном состоянии. В некоторых из таких клеток первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, присутствуют стабильно в агрегированном состоянии.
[0046] В другом аспекте представлены биосенсорные клетки SAM-тау и популяция таких клеток, например, популяция из одной или большего числа клеток, содержащих химерный белок Cas, который содержит белок Cas с инактивированной нуклеазной активностью (т.е. каталитически неактивный белок Cas), слитый с одним или большим числом доменов активации транскрипции, химерный адаптерный белок, который содержит адаптерный белок, слитый с одним или большим числом доменов активации транскрипции, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером.
[0047] В некоторых из таких клеток первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка представляют собой содержащий повторы домен тау-белка человека. В некоторых из таких клеток первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат мутацию, способствующую агрегации. Необязательно, первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат мутацию тау-белка P301S. В некоторых из таких клеток первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат домен тау-белка с четырьмя повторами. В некоторых из таких клеток первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка содержат последовательность с SEQ ID NO: 11. В некоторых из таких клеток первый содержащий повторы домен тау-белка и второй содержащий повторы домен тау-белка являются одинаковыми. В некоторых из таких клеток первый содержащий повторы домен тау-белка и второй содержащий повторы домен тау-белка являются одинаковыми, и каждый из них содержит домен тау-белка с четырьмя повторами, содержащий мутацию тау-белка P301S.
[0048] В некоторых из таких клеток первый репортер и второй репортер представляют собой флуоресцентные белки. Необязательно, первый репортер и второй репортер представляют собой пару с резонансным переносом энергии флуоресценции (FRET). Необязательно, первый репортер представляет собой голубой флуоресцентный белок (CFP), а второй репортер представляет собой желтый флуоресцентный белок (YFP).
[0049] В некоторых из таких клеток белок Cas представляет собой белок Cas9. Необязательно, белок Cas представляет собой Cas9 из Streptococcus pyogenes. В некоторых из таких клеток химерный белок Cas содержит белок Cas с инактивированной нуклеазной активностью, слитый с доменом активации транскрипции VP64, необязательно при этом указанный химерный белок Cas содержит, в направлении от N-конца до С-конца: белок Cas с инактивированной нуклеазной активностью; сигнал ядерной локализации; и домен активатора транскрипции VP64. В некоторых из таких клеток адаптерный белок представляет собой белок оболочки MS2, а один или большее число доменов активации транскрипции в химерном адаптерном белке содержат домен активации транскрипции р65 и домен активации транскрипции HSF1, необязательно при этом указанный химерный адаптерный белок содержит, в направлении отТчГ-конца до С-конца: белок оболочки MS2; сигнал ядерной локализации; домен активации транскрипции р65; и домен активации транскрипции HSF1. В некоторых из таких клеток химерный белок Cas содержит последовательность с SEQ ID NO: 36, необязательно при этом указанный химерный белок Cas кодируется кодирующей последовательностью, содержащей последовательность, указанную в SEQ ID NO: 38. В некоторых из таких клеток химерный адаптерный белок содержит последовательность с SEQ ID NO: 37, необязательно при этом указанный химерный адаптерный белок кодируется кодирующей последовательностью, содержащей последовательность, указанную в SEQ ID NO: 39.
[0050] В некоторых из таких клеток химерный белок Cas, химерный адаптерный белок, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, стабильно экспрессируются в указанной клетке. В некоторых из таких клеток нуклеиновые кислоты, кодирующие химерный белок Cas, химерный адаптерный белок, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, геномно интегрированы в указанную клетку.
[0051] Некоторые из таких клеток представляют собой эукариотические клетки. Необязательно, клетки представляют собой клетки млекопитающих. Необязательно, клетки представляют собой клетки человека. Необязательно, клетки представляют собой клетки НЕК293Т. Необязательно, клетки находятся in vitro.
[0052] В некоторых из таких клеток первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, не присутствуют стабильно в агрегированном состоянии. В некоторых из таких клеток первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, присутствуют стабильно в агрегированном состоянии.
[0053] В другом аспекте представлены культуры in vitro, содержащие любую из вышеуказанных популяций клеток или любую из клеток, описанных в данном документе, и культуральную среду.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
[0054] На фиг. 1 (не в масштабе) представлено схематическое изображение изоформы тау-белка 2N4R. Линии тау-биосенсоров включают в себя только тау-4RD-YFP и тау-4RD-CFP в качестве трансгенов, но не полный 2N4R.
[0055] На фиг. 2 представлено схематическое изображение того, как образование агрегатов отслеживается с помощью резонансного переноса энергии флуоресценции (FRET) в линиях клеток-биосенсоров тау-белка. Белок тау4RD-CFP возбуждается фиолетовым светом и излучает синий свет. Слитый белок тау4RD-YFP возбуждается синим светом и излучает желтый свет. Если агрегации нет, то возбуждение фиолетовым светом не приведет к FRET. Если агрегация тау-белка есть, то возбуждение фиолетовым светом приведет к FRET и излучению желтого света.
[0056] На фиг. 3А показана относительная экспрессия иРНК Cas9 в клонах биосенсорных клеток тay4RD-CFP/тay4RD-YFP (TCY), трансдуцированных лентивирусными экспрессионными конструкциями для Cas9, по сравнению с клоном Cas9Hl, который представляет собой ранее выделенный контрольный клон TCY с недостаточной экспрессией Cas9.
[0057] На фиг. 3В показана эффективность разрезания покуса PERK и локуса SNCA в клонах TCY Cas9 через трое и семеро суток после трансдукции с использованием единых гидовых РНК (егРНК, англ. «sgRNA»), нацеленных па PERK и SNCA, соответственно.
[0058] На фиг. 4 представлено схематическое изображение стратегии нарушения целевых генов в биосенсорной клетке TCY Cas9 с использованием полногеномной библиотеки егРНК CRISPR/Cas9.
[0059] На фиг. 5 представлено схематическое изображение получения субклонов тау4RD-CFP/тау4RD-YFP/Cas9 Аг[+], содержащих стабильно размножающиеся агрегаты тау-белка. Также представлено изображение, полученное с помощью FRET-микроскопии, на котором показан субклон с агрегатами тау-белка.
[0060] На фиг. 6 представлено схематическое изображение эксперимента по оценке генов, нарушенных агрегацией тау-белка.
[0061] На фиг. 7 представлены результаты неконтролируемой кластеризации, которые показывают, что образцы клонов Аг[+] и образцы клонов Аг[-] различаются по статусу агрегации (процедура кластеризации: логарифмическая трансформация считываний на миллион т.п.н. (RPKM); метрика расстояния: 1 - абсолютное значение коэффициента корреляции Пирсона; иерархическая кластеризация).
[0062] На фиг. 8 представлены гены, значительно нарушенные агрегацией в клетках Тау Аг[+] (значительно нарушенные гены, определяемые как кратность изменения ≥1,5 (повышение или понижение) и значение р≤0,01.
[0063] На фиг. 9 представлена обобщенная схема объединенного полногеномного скрининга нуклеаз CRISPR (CRISPRn) для выявления генетических уязвимостей, связанных с агрегацией тау-белка.
[0064] На фиг. 10А представлена обобщенная схема идентификации основных генов (егРНК, нацеленных на те гены, которые истощены как в Аг[+], так и в Аг[-]).
[0065] На фиг. 10 В представлена обобщенная схема идентификации синтетических летальных генов (егРНК, нацеленные на те гены, которые истощены в Аг[+], но не в Аг[-]).
[0066] На фиг. 11 представлена схема исследования полной временной динамики для идентификации генов, для которых существует паттерн истощения егРНК с течением времени.
[0067] На фиг. 12 показано число генов, для которых егРНК были истощены в образцах Аг[+] и Аг[-].
[0068] На фиг. 13 представлен график, показывающий перекрытие основных генов, идентифицированных в биосенсорном тау-скрининге, по сравнению с другими опубликованными литературными источниками.
[0069] На фиг. 14 представлен график, показывающий идентификацию 71 гена с однозначно истощенными егРНК с течением времени по сравнению с 3-ми сутками в образцах Аг[+] по сравнению с образцами Аг[-] в трех повторных экспериментах.
[0070] На фиг. 15А и 15В показано кратное изменение во времени (истощение егРНК) по сравнению с 3-ми сутками в клетках Аг[+] и Аг[-] для егРНК, нацеленных на три основных гена-кандидата (основные гены 6, 8 и 9 (ОГ6, ОГ8, и ОГ9); фиг. 15А) и три кандидатных синтетических летальных гена (целевые гены 1, 5 и 6 (ЦГ1, ЦГ5 и ЦГ6); фиг. 15 В).
[0071] На фиг. 16 показано распределение числа считываний специальной библиотеки егРНК, состоящей из 462 уникальных гидовых егРНК, нацеленных на 71 предполагаемый синтетический летальный ген (для которых егРНК были выборочно истощены в клетках Аг[+] во время первичного скрининга) и 10 предполагаемых основных генов (для которых егРНК были истощены как в клетках Аг[+], так и в клетках Аг[-] во время первичного скрининга).
[0072] На фиг. 17 показано распределение числа считываний библиотеки егРНК, состоящей из 500 уникальных ненацеленных контрольных егРНК (неспецифических егРНК, которые не изменялись с течением времени в первичном скрининге).
[0073] На фиг. 18 представлена обобщенная схема вторичного скрининга для валидации 71 предполагаемого синтетического летального гена и 10 предполагаемых основных генов с использованием специальных библиотек егРНК.
[0074] На фиг. 19А - 19С показано кратное изменение во времени (истощение егРНК) по сравнению с 3-ми сутками в клетках Аг[+] и Аг[-] для егРНК, нацеленных на десять основных генов-кандидатов (ОГ1 - ОГ10) во вторичном скрининге.
[0075] На фиг. 20 представлен пример графика, показывающего различимый паттерн истощения егРНК между клетками Аг[+] и клетками Аг[-] (т.е. более резкое истощение клеток Аг[+]).
[0076] На фиг. 21 представлена обобщенная схема идентификации синтетических летальных генов во вторичном скрининге путем выявления егРНК, которые демонстрируют как значимое значение р для динамики с течением времени, так и паттерн истощения в каждом эксперименте (10-е сутки <3-й сутки, 14-е сутки <7-е сутки и 17-е сутки <10-е сутки).
[0077] На фиг. 22А показано кратное изменение во времени (истощение егРНК) в клетках Аг[+] и Аг[-] для шести егРНК, нацеленных на целевой ген 1 (ЦГ1), и для шести егРНК, нацеленных на целевой ген 2 (ЦГ2).
[0078] На фиг. 22 В показано кратное изменение во времени (истощение егРНК) в клетках Аг[+] и Аг[-] для шести егРНК, нацеленных на целевой ген 3 (ЦГ3), и для шести егРНК, нацеленных на целевой ген 4 (ЦГ4).
[0079] На фиг. 23 представлена обобщенная схема идентификации синтетических летальных генов во вторичном скрининге путем выявления CRISPRa егРНК, которые демонстрируют как значимое значение р для динамики с течением времени, так и паттерн истощения в каждом эксперименте (10-е сутки <3-й сутки, 14-е сутки <7-е сутки и 17-е сутки <10-е сутки).
[0080] На фиг. 24А и фиг. 24 В показано кратное изменение во времени (истощение егРНК) в клетках Аг[+] и Аг[-] для егРНК, нацеленных на целевые гены 7-15 (ЦГ7 - ЦП 5).
[0081] На фиг. 25 показана схема дополнительной валидации целевых генов 1, 2 и 4 с использованием метода проточной цитометрии для количественного определения жизнеспособности клеток и гибели клеток для популяций клеток Аг[+] и клеток Аг[-] после трансдукции лентивирусными векторами, доставляющими егРНК, нацеленные на целевые гены 1, 2 и 4.
[0082] На фиг. 26 показаны жизнеспособность клеток и гибель клеток для популяций клеток Аг[+] и клеток Аг[-] после трансдукции лентивирусными векторами, доставляющими егРНК, нацеленные на целевые гены 1, 2 и 4.
ОПРЕДЕЛЕНИЯ
[0083] Термины «белок», «полипептид» и «пептид», используемые в контексте данного документа взаимозаменяемо, включают в себя полимерные формы любой длины, состоящие из аминокислот, включая кодируемые и некодируемые аминокислоты, и химически или биохимически модифицированные либо дериватизированные аминокислоты. Данные термины также включают в себя модифицированные полимеры, такие как полипептиды, имеющие модифицированные пептидные каркасы. Термин «домен» относится к любой части белка или полипептида, имеющей конкретную функцию или структуру.
[0084] Указывают, что у белков есть «N-конец» и «С-конец». Термин «N-конец» относится к началу белка или полипептида, оканчивающемуся аминокислотой со свободной аминогруппой (-NH2). Термин «С-конец» относится к концу аминокислотной цепи (белка или полипептида), оканчивающейся свободной карбоксильной группой (-СООН).
[0085] Термины «нуклеиновая кислота» и «полинуклеотид», используемые в контексте данного документа взаимозаменяемо, включают в себя полимерные формы любой длины, состоящие из нуклеотидов, включая рибонуклеотиды, дезоксирибонуклеотиды или их аналоги либо их модифицированные версии. Они включают в себя одно-, двух- и многоцепочечные ДНК или РНК, геномные ДНК, кДНК, гибриды ДНК РНК и полимеры, содержащие пуриновые основания, пиримидиновые основания или другие встречающиеся в природе, химически модифицированные, биохимически модифицированные, не встречающиеся в природе или дериватизированные нуклеотидные основания.
[0086] Указывают, что нуклеиновые кислоты имеют «5'-концы» и «3'-концы», потому что мононуклеотиды реагируют с образованием олигонуклеотидов таким образом, что 5'-фосфат одного пентозного кольца мононуклеотида присоединяется к 3'-кислороду соседнего мононуклеотида в одном направлении через фосфодиэфирную связь. Конец олигонуклеотида называется «5'-концом», если его 5'-фосфат не связан с З'-кислородом пентозного кольца мононуклеотида. Конец олигонуклеотида называется «3'-концом», если его 3'-кислород не связан с 5'-фосфатом другого пентозного кольца мононуклеотида. Можно сказать, что последовательность нуклеиновой кислоты, даже если она находится внутри по отношению к более крупному олигонуклеотиду, имеет 5'- и 3'-концы. Либо в линейной, либо в кольцевой молекуле ДНК дискретные элементы упоминаются как «расположенные в направлении к 5'» или «расположенные в направлении к 3'».
[0087] Термин «геномно интегрированный» относится к нуклеиновой кислоте, которая была введена в клетку таким образом, что данная нуклеотидная последовательность интегрировалась в геном данной клетки. Для стабильного включения нуклеиновой кислоты в геном клетки можно применять любой протокол.
[0088] Термин «нацеливающий вектор» относится к рекомбинантной нуклеиновой кислоте, которая может быть введена в целевое положение в геноме клетки путем гомологичной рекомбинации, лигирования, опосредованного негомологичным соединением концов, или любым другим способом рекомбинации.
[0089] Термин «вирусный вектор» относится к рекомбинантной нуклеиновой кислоте, которая включает в себя по меньшей мере один элемент вирусного происхождения и включает в себя элементы, достаточные для упаковки в частицу вирусного вектора или допускающие указанную упаковку. Вектор и (или) частица могут быть использованы с целью переноса ДНК, РНК или других нуклеиновых кислот в клетки либо ex vivo, либо in vivo. Известны многочисленные формы вирусных векторов.
[0090] Термин «дикий тип» включает в себя реально существующие объекты, имеющие структуру и (или) активность, как обнаруживается в нормальном (в отличие от мутантного, болезненного, измененного и т.д.) состоянии или контексте. Гены и полипептиды дикого типа часто существуют в нескольких различных формах (например, аллелях).
[0091] Термин «эндогенная последовательность» относится к последовательности нуклеиновой кислоты, которая встречается в природе внутри клетки или организма. Например, эндогенная последовательность МАРТ клетки или организма относится к нативной последовательности МАРТ, которая естественным образом встречается в локусе МАРТ внутри клетки или организма.
[0092] «Экзогенные» молекулы или последовательности включают в себя молекулы или последовательности, которые обычно не присутствуют в клетке в данной форме. Нормальное присутствие включает в себя присутствие в зависимости от конкретной стадии развития и условий окружающей среды клетки. Экзогенная молекула или последовательность, например, может включать в себя мутированную версию соответствующей эндогенной последовательности внутри клетки, такую как гуманизированная версия эндогенной последовательности, или может включать в себя последовательность, соответствующую эндогенной последовательности внутри клетки, но в другой форма (т.е. не в хромосоме). Напротив, эндогенные молекулы или последовательности включают в себя молекулы или последовательности, которые обычно присутствуют в данной форме в конкретной клетке на конкретной стадии развития в определенных условиях окружающей среды.
[0093] Термин «гетерологичный» при использовании в контексте нуклеиновой кислоты или белка, указывает на то, что данная нуклеиновая кислота или данный белок содержат по меньшей мере два сегмента, которые в природе не встречаются вместе в одной и той же молекуле. Например, термин «гетерологичный», когда он используется в отношении сегментов нуклеиновой кислоты или сегментов белка, указывает, что данная нуклеиновая кислота или данный белок содержат две или большее число субпоследовательностей, которые не находятся в такой взаимосвязи друг с другом (например, соединенные вместе) в природе. В качестве одного примера, «гетерологичная» область вектора нуклеиновой кислоты представляет собой сегмент нуклеиновой кислоты внутри молекулы или сегмент присоединенный к другой молекуле нуклеиновой кислоты, который не обнаруживается в ассоциации с другой молекулой в природе. Например, гетерологичная область вектора нуклеиновой кислоты может включать в себя кодирующую последовательность, фланкированную последовательностями, не обнаруживаемыми в ассоциации с указанной кодирующей последовательностью в природе. Аналогичным образом, «гетерологичная» область белка представляет собой сегмент из аминокислот внутри другой молекулы белка или присоединенный к другой молекуле белка, не обнаруживаемый в ассоциации с указанной другой молекулой белка в природе (например, слитый белок или белок с меткой). Подобным образом, нуклеиновая кислота или белок могут содержать гетерологичную метку или гетерологичную последовательность секреции или локализации.
[0094] Термин «локус» относится к конкретному местоположению гена (или значимой последовательности), последовательности ДНК, последовательности, кодирующей полипептид, или положению на хромосоме генома организма. Например, «локус МАРТ» может относиться к определенному местоположению гена МАРТ, последовательность ДНК МАРТ, последовательности, кодирующей ассоциированный с микротрубочками тау-белок, или положению МАРТ на хромосоме генома организма, который был идентифицирован как такой, в котором находится такая последовательность. «Локус МАРТ» может содержать регуляторный элемент гена МАРТ, включая, например, энхансер, промотор, 5'- и (или) 3'-нетранслируемую область (НТО, англ. «UTR»), или их комбинацию.
[0095] Термин «ген» относится к последовательности ДНК в хромосоме, которая кодирует продукт (например, РНК-продукт и (или) продукт-полипептид) и включает в себя кодирующую область, прерванную некодирующими нитронами, и последовательность, расположенную смежно с указанной кодирующей областью как на 5'-конце, так и на 3'-конце, таким образом, что ген соответствует полноразмерной иРНК (включая 5'- и 3' не транслируемые последовательности). Термин «ген» также включает в себя другие некодирующие последовательности, включая регуляторные последовательности (например, промоторы, энхансеры и сайты связывания факторов транскрипции), сигналы полиаденилирования, участок внутренней посадки рибосомы, сайленсеры, изолирующие последовательности и области прикрепления к матриксу. Эти последовательности могут быть расположены вблизи кодирующей области гена (например, в пределах 10 т.п.о.) или в отдаленных сайтах, и они влияют на уровень или скорость транскрипции и трансляции данного гена.
[0096] Термин «аллель» относится к вариантной форме гена. Некоторые гены имеют множество различных форм, которые расположены в одной и той же позиции, или генетическом локусе, на хромосоме. Диплоидный организм имеет два аллеля в каждом генетическом локусе. Каждая пара аллелей отражает собой генотип определенного генетического локуса. Генотипы описываются как гомозиготные, если есть два идентичных аллеля в определенном локусе, и как гетерозиготные, если эти два аллеля различаются.
[0097] «Промотор» представляет собой регуляторную область ДНК, обычно содержащую ТАТА-бокс, которая способна направлять РНК-полимеразу II с целью инициации синтеза РНК в соответствующем сайте инициации транскрипции для конкретной полинуклеотидной последовательности. Промотор может дополнительно включать в себя другие области, которые влияют на скорость инициации транскрипции. Описанные в данном документе промоторные последовательности модулируют транскрипцию функционально связанного полинуклеотида. Промотор может проявлять активность в одном или нескольких типах клеток, описанных в данном документе (например, в клетке человека, плюрипотентной клетке, эмбрионе на одноклеточной стадии, дифференцированной клетке или их комбинации). Промотор может представлять собой, например, конститутивно активный промотор, условный промотор, индуцибельный промотор, ограниченный во времени промотор (например, промотор, регулируемый развитием) или ограниченный в пространстве промотор (например, клеточно-специфический или тканеспецифический промотор). Примеры промоторов можно найти, например, в патентном документе WO 2013/176772, который включен в данный документ посредством ссылки во всей своей полноте для всех целей.
[0098] Термин «функциональная связь» или «функционально связанный (-ая, -ые)» включает в себя контактное расположение двух или большего числа компонентов (например, промотора и другого элемента последовательности), таким образом, что оба компонента функционируют нормально и допускают возможность того, что по меньшей мере один из данных компонентов может опосредовать функцию, которая воздействует на по меньшей мере один из других компонентов. Например, промотор может быть функционально связан с кодирующей последовательностью, если промотор контролирует уровень транскрипции кодирующей последовательности в ответ на присутствие или отсутствие одного либо большего числа факторов регуляции транскрипции. Функциональная связь может включать в себя случаи, когда такие последовательности расположены смежно друг с другом или действуют в транс-положении (например, регуляторная последовательность может действовать на расстоянии, чтобы контролировать транскрипцию кодирующей последовательности).
[0099] Термин «вариант» относится к нуклеотидной последовательности, отличающейся от последовательности, наиболее распространенной в популяции (например, на один нуклеотид), или к последовательности белка, отличающейся от последовательности, наиболее распространенной в популяции (например, на одну аминокислоту).
[00100] Термин «фрагмент» по отношению к белку означает белок, который имеет меньшую длину или содержит меньшее число аминокислот, чем полноразмерный белок. Термин «фрагмент» по отношению к нуклеиновой кислоте означает нуклеиновую кислоту, которая имеет меньшую длину или содержит меньшее число нуклеотидов, чем полноразмерная нуклеиновая кислота. Фрагмент может представлять собой, например, N-концевой фрагмент (т.е. удалена С-концевая часть белка), С-концевой фрагмент (т.е. удалена N-концевая часть белка) или внутренний фрагмент.
[00101] «Идентичность последовательностей», или «идентичность», в контексте двух полинуклеотидных или полипептидных последовательностей относится к остаткам в двух последовательностях, которые являются одинаковыми при выравнивании последовательностей для максимального соответствия в указанном окне сравнения. Когда процент идентичности последовательностей используется в отношении белков, тогда положения остатков, которые не идентичны, часто отличаются по консервативным аминокислотным заменам, в которых аминокислотные остатки заменены на другие аминокислотные остатки с аналогичными химическими свойствами (например, заряд или гидрофобность) и, следовательно, не меняют функциональных свойств данной молекулы. Когда последовательности отличаются по консервативным аминокислотным заменам, процент идентичности последовательностей может быть увеличен для корректировки относительно консервативного характера замены. Указывают, что последовательности, отличающиеся по таким консервативным заменам, имеют «сходство последовательностей», или «сходство». Средства для выполнения данной корректировки хорошо известны. Обычно это включает в себя оценку консервативной замены как частичного, а не полного несоответствия, что тем самым увеличивает процент идентичности последовательностей. Таким образом, например, если идентичной аминокислоте присваивается оценка, равная 1, а неконсервативной замене присваивается оценка, равная нулю, то консервативной замене присваивается оценка, равная от нуля до 1. Оценка консервативных замен рассчитывается, например, так, как реализовано в программе PC/GENE (bitelligenetics, Маунтин-Вью, Калифорния, США).
[00102] «Процент идентичности последовательностей» включает в себя значение, определенное путем сравнения двух оптимально выровненных последовательностей (наибольшее количество полностью совпадающих остатков) в окне сравнения, где часть полинуклеотидной последовательности в окне сравнения может содержать добавления или делеции (т.е. гэпы) по сравнению с референсной последовательностью (которая не содержит добавлений или делеций) для оптимального выравнивания данных двух
последовательностей. Данный процент рассчитывается путем определения числа положений, в которых одно и то же основание нуклеиновой кислоты или один и тот же аминокислотный остаток встречается в обеих последовательностях, чтобы получить количество совпадающих положений, с последующим делением числа совпадающих положений на общее число положений в окне сравнения и умножением полученного результата на 100, чтобы получить процент идентичности последовательностей. Если не указано иное (например, более короткая последовательность включает в себя связанную гетерологичную последовательность), окно сравнения представляет собой полную протяженность более короткой из двух сравниваемых последовательностей.
[00103] Если не указано иное, значения идентичности / сходства последовательностей включает в себя значение, полученное с использованием программного обеспечения GAP версии 10 с использованием следующих параметров: % идентичности и % сходства для нуклеотидной последовательности с использованием штрафа в 50 за открытие гэпа и штрафа в 3 за удлинение гэпа, а также оценочной матрицы nwsgapdna.cmp; % идентичности и % сходства для нуклеотидной последовательности с использованием штрафа в 8 за открытие гэпа и штрафа в 2 за удлинение гэпа, а также оценочной матрицы BLOSUM62; или с помощью любой эквивалентной программы. «Эквивалентная программа» включает в себя любую программу для сравнения последовательностей, которая для любых двух рассматриваемых последовательностей генерирует выравнивание, имеющее идентичные совпадения нуклеотидных или аминокислотных остатков и идентичный процент идентичности последовательностей по сравнению с соответствующим выравниванием, созданным в программе GAP версии 10.
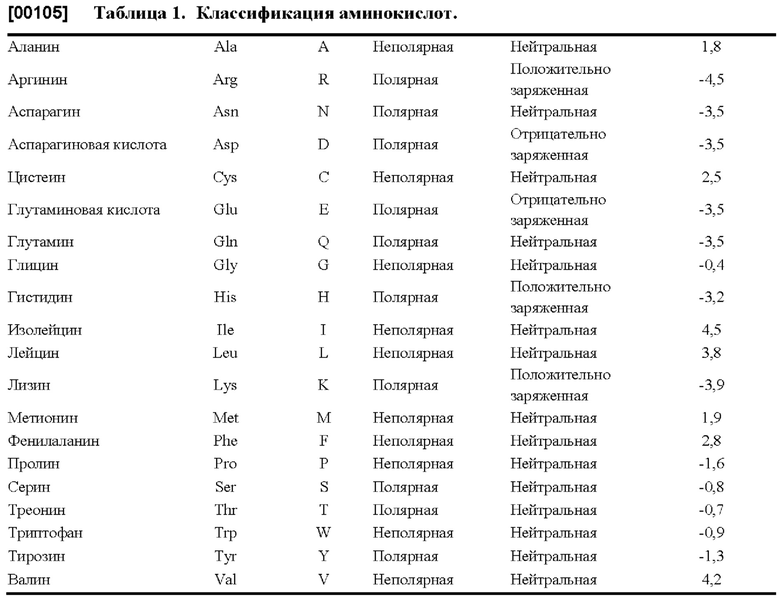
[00104] Термин «консервативная аминокислотная замена» относится к замене аминокислоты, которая обычно присутствует в данной последовательности, на другую аминокислоту аналогичного размера, заряда или полярности. Примеры консервативных замен включают в себя замену неполярного (гидрофобного) остатка, такого как изолейцин, валин или лейцин, на другой неполярный остаток. Аналогичным образом, примеры консервативных замен включают в себя замену одного полярного (гидрофильного) остатка на другой, например, замену аргинина на лизин и наоборот, замену глутамина на аспарагин и наоборот или замену глицина на серии и наоборот. Кроме того, замена основного остатка, такого как лизин, аргинин или гистидин, на другой остаток, или замена одного кислотного остатка, такого как аспарагиновая кислота или глутаминовая кислота, на другой кислотный остаток представляют собой дополнительные примеры консервативных замен. Примеры неконсервативных замен включают в себя замену неполярного (гидрофобного) аминокислотного остатка, такого как изолейцин, валин, лейцин, аланин или метионин, на полярный (гидрофильный) остаток, такой как цистеин, глутамин, глутаминовая кислота или лизин, и (или) замену полярного остатка на неполярный остаток. Типовая классификация аминокислот обобщенно приведена ниже.
[00106] Термин «гомологичная» последовательность (например, последовательность нуклеиновой кислоты) включает в себя последовательность, которая либо идентична, либо по существу сходна с известной референсной последовательностью таким образом, что она, например, по меньшей мере на 50%, по меньшей мере на 55%, по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична известной референсной последовательности. Гомологичные последовательности могут включать в себя, например, ортологичную последовательность и паралогичные последовательности. Гомологичные гены, например, обычно происходят от общей предковой последовательности ДНК либо в результате видообразующего события (ортологичные гены), либо в результате события генетической дупликации (паралогичные гены). «Ортологичные» гены включают в себя гены разных видов, которые произошли от общего предкового гена путем видообразования. Ортологи обычно сохраняют одну и ту же функцию в ходе эволюции. «Паралогичные» гены включают в себя гены, связанные посредством дупликации в геноме. Паралоги могут развить новые функции в ходе эволюции.
[00107] Термин «in vitro» включает в себя искусственную среду и процессы либо реакции, которые происходят в искусственной среде (например, в пробирке, в выделенной клетке или клеточной линии). Термин «in vivo» включает в себя естественную среду (например, клетку, или организм, или тело) и процессы либо реакции, которые происходят в естественной среде. Термин «ех vivo» включает в себя клетки, которые были удалены из организма индивида, а также процессы или реакции, которые происходят в таких клетках.
[00108] Термин «репортерный ген» относится к нуклеиновой кислоте, имеющей последовательность, кодирующую продукт гена (обычно фермент), которая поддается легкому и количественному анализу, когда конструкция, содержащая последовательность репортерного гена, функционально связанную с гетерологичный промотором и (или) энхансерным элементом, вводится в клетки, которые содержат (или которые могут быть получены так, чтобы они содержали) факторы, необходимые для активации промоторных и (или) энхансерных элементов. Примеры репортерных генов включают в себя, но не ограничиваются ими, гены, кодирующие бета-галактозидазу (lacZ), гены бактериальной хлорамфениколацетилтрансферазы (cat), гены люциферазы светлячка, гены, кодирующие бета-глюкуронидазу (GUS), и гены, кодирующие флуоресцентные белки. «Репортерный белок» относится к белку, кодируемому репортерным геном.
[00109] Термин «флуоресцентный репортерный белок», используемый в контексте данного документа, означает репортерный белок, который определяется на основе флуоресценции, при этом флуоресценция может исходить либо непосредственно от репортерного белка, либо от эффекта репортерного белка на флуорогенный субстрат, либо от белка с аффинностью к связыванию с флуоресцентно меченным соединением. Примеры флуоресцентных белков включают в себя зеленые флуоресцентные белки (например, GFP, GFP-2, tagGFP, turboGFP, eGFP, Emerald, Azami Green, мономерный Azami Green, CopGFP, AceGFP и ZsGreenl), желтые флуоресцентные белки (например, YFP, eYFP, Citrine, Venus, YPet, PhiYFP и ZsYellowl), синие флуоресцентные белки (например, BFP, eBFP, eBFP2, Azurite, mKalamal, GFPuv, Sapphire и T-sapphire), голубые флуоресцентные белки (например, CFP, eCFP, Cerulean, CyPet, AmCyanl и Midoriishi-Cyan), красные флуоресцентные белки (например, RFP, mKate, mKate2, mPlum, мономер DsRed, mCherry, mRFPl, DsRed-Express, DsRed2, мономер DsRed, тандем HcRed, HcRedl, AsRed2, eqFP611, mRaspberry, mStrawberry и Jred), оранжевые флуоресцентные белки (например, mOrange, mKO, Kusabira-Orange, мономерный Kusabira-Orange, mTangerine и tdTomato) и любой другой подходящий флуоресцентный белок, присутствие которого в клетках можно определить с помощью способов проточной цитометрии.
[00110] Репарация в ответ на двухцепочечные разрывы (ДЦР, англ. «DSB») происходит главным образом посредством двух консервативных путей репарации ДНК: гомологичная рекомбинация (ГР, англ. «HR») и негомологичное соединение концов (НГСК, англ. «NHEJ»). См. работу Kasparek & Humphrey (2011) Seminars in Cell & Dev. Biol. 22: 886 - 897, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей. Точно так же репарация целевой нуклеиновой кислоты, опосредованная экзогенной донорной нуклеиновой кислотой, может включать в себя любой процесс обмена генетической информацией между двумя указанными полинуклеотидами.
[00111] Термин «рекомбинация» включает в себя любой процесс обмена генетической информацией между двумя полинуклеотидами и может происходить по любому механизму. Рекомбинация может происходить посредством гомологически направленной репарации (ГНР, англ. «HDR») или гомологичной рекомбинации (ГР). ГНР или ГР включает в себя вид репарации нуклеиновой кислоты, который может требовать гомологии нуклеотидной последовательности, использует «донорную» молекулу в качестве матрицы для репарации «целевой» молекулы (т.е. той, в которой произошел двух цепочечный разрыв) и приводит к передаче генетической информации от донора к целевой молекуле. Не желая быть связанными какой-либо конкретной теорией, такой перенос может включать в себя исправление ошибки спаривания нуклеотидов гетеродуплексной ДНК, которая образуется между поврежденной целевой молекулой и донором, и (или) зависимый от синтеза отжиг цепи, при котором донор используется для повторного синтеза генетической информации, которая станет частью целевой молекулы, и (или) связанные процессы. В некоторых случаях донорный полинуклеотид, часть донорного полинуклеотида, копия донорного полинуклеотида или часть копии донорного полинуклеотида интегрируется в целевую ДНК. См. работы Wang et al. (2013) Cell 153: 910-918; Mandalos et al. (2012) PLOS ONE 7: e45768: 1-9; и Wang et al. (2013) NatBiotechnol. 31: 530-532, каждая из которых включена в данный документ посредством ссылки во всей своей полноте для всех целей.
[00112] НГСК включает в себя репарацию двух цепочечных разрывов в нуклеиновой кислоте путем прямого лигирования концов данных разрывов друг с другом или с экзогенной последовательностью без необходимости в гомологичной матрице. Лигирование несмежных последовательностей с помощью НГСК часто может приводить к делециям, вставкам или транслокациям вблизи сайта двухцепочечного разрыва. Например, НГСК может также привести к нацеленной интеграции экзогенной донорной нуклеиновой кислоты посредством прямого лигирования концов разрывов с концами указанной экзогенной донорной нуклеиновой кислоты (т.е. захвату на основе НГСК). Такая нацеленная интеграция, опосредованная НГСК, может быть предпочтительной для вставки экзогенной донорной нуклеиновой кислоты, когда пути гомологически направленной репарации (ГНР) не могут быть легко использованы (например, в неделящихся клетках, первичных клетках и клетках, в которых плохо осуществляется репарация ДНК на основе гомологии). В дополнение к этому, в отличие от гомологически направленной репарации, не требуется знание больших областей идентичности последовательностей, фланкирующих сайт расщепления, что может быть полезно при попытке направленной вставки в организмы, о геномной последовательности геномов которых имеется лишь ограниченное знание. Интеграция может происходить посредством лигирования тупых концов между экзогенной донорной нуклеиновой кислотой и расщепленной геномной последовательностью или посредством лигирования липких концов (т.е. концов, имеющих 5'- или З'-выступы) с использованием экзогенной донорной нуклеиновой кислоты, фланкированной выступами, которые совместимы с теми, которые генерируются нуклеазным агентом в указанной расщепленной геномной последовательности. См., например, US 2011/020722, WO 2014/033644, WO 2014/089290 и Maresca et al. (2013) Genome Res. 23 (3): 539 - 546, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. Если лигированы тупые концы, может потребоваться резекция целевой последовательности и (или) донорной последовательности для создания областей микрогомологии, необходимых для соединения фрагментов, что может создать нежелательные изменения в указанной целевой последовательности.
[00113] Композиции или способы, «содержащие» или «включающие в себя» один или большее число из перечисленных элементов, могут включать в себя другие элементы, не перечисленные специально. Например, композиция, которая «содержит» или «включает в себя» белок, может содержать белок отдельно или в комбинации с другими ингредиентами. Переходная фраза «состоящий (-ая, -ие) по существу из» означает, что объем формулы изобретения следует интерпретировать как включающий в себя указанные в данной формуле изобретения элементы, а также те элементы, которые не оказывают существенное влияние на основную (-ые) и новую (-ые) характеристику (-и) заявленного изобретения. Таким образом, термин «состоящий (-ая, -ие) по существу из» при использовании в формуле данного изобретения не предназначен для интерпретации как эквивалентный термину «содержащий (-ая, -ие)».
[00114] «Необязательный» или «необязательно» означает, что описанное далее событие или обстоятельство может или не может произойти, и что данное описание включает в себя случаи, в которых данное событие или данное обстоятельство происходит, и случаи, когда данное событие или данное обстоятельство не происходит.
[00115] Обозначение диапазона значений включает в себя все целые числа в пределах данного диапазона или все целые числа, определяющие данный диапазон, а также все поддиапазоны, определяемые целыми числами в пределах данного диапазона.
[00116] Если иное явным образом не следует из контекста, термин «около» охватывает значения в пределах стандартной погрешности измерения (например, стандартной ошибки среднего - СОС, англ. «SEM») для указанного значения.
[00117] Термин «и (или)» относится к и охватывает любые и все возможные комбинации одного или большего числа связанных перечисленных элементов, а также отсутствие комбинаций в случае интерпретации как альтернативы («или»).
[00118] Термин «или» относится к любому одному члену конкретного перечня, а также включает в себя любую комбинацию элементов данного перечня.
[00119] Формы единственного числа включают в себя соответствующие им формы множественного числа, если из контекста явным образом не следует иное. Например, термины «белок» или «по меньшей мере один белок» могут включать в себя множество белков, включая их смеси.
[00120] «Статистически значимое» означает р≤0,05.
ПОДРОБНОЕ ОПИСАНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
I. Обзор
[00121] Представлены готовые к Cas-белку клетки-биосенсоры тау-белка, а также способы получения и применения таких клеток для скрининга в отношении генетической уязвимости, связанной с агрегацией тау-белка. Также представлены готовые к медиатору синергической активации (SAM) CRISPR/Cas клетки-биосенсоры тау-белка, а также способы получения и применения таких клеток для скрининга в отношении генетической уязвимости, связанной с агрегацией тау-белка.
[00122] Чтобы идентифицировать гены и пути, которые проявляют синтетическую летальность с ассоциированными с заболеванием белковыми агрегатами, была разработана платформа для проведения полногеномных скринингов с использованием библиотек егРНК нуклеаз CRISPR (CRISPRn). Указанная платформа идентифицирует гены, нарушение которых вызывает гибель клеток, особенно в контексте аномальной агрегации белка. Чтобы идентифицировать гены и пути, которые проявляют синтетическую летальность с ассоциированными с заболеванием белковыми агрегатами, была разработана платформа для проведения полногеномных скринингов с использованием библиотек егРНК активации CRISPR (CRISPRa). Указанная платформа идентифицирует гены, активация которых вызывает гибель клеток, особенно в контексте аномальной агрегации белка. Идентификация таких генов может пролить свет на механизмы нейротоксичности, связанной с агрегатами, и генетические пути, которые способствуют гибели нейронов в контексте нейродегенеративного заболевания.
[00123] В указанных скринингах применяется линия клеток-биосенсоров тау-белка (например, линия клеток человека или НЕК293Т), состоящая из клеток, стабильно экспрессирующих содержащий повторы домен тау-белка (например, домен тау-белка, содержащий четыре повтора - тау_4RD) с патогенной мутацией (например, патогенной мутацией P301S), связанный с уникальными репортерами, которые могут действовать вместе как внутриклеточный биосенсор, который при агрегировании производит обнаруживаемый сигнал. В одном неограничивающем примере клеточные линии содержат два трансгена, стабильно экспрессирующих варианты ассоциированного с заболеванием белка, слитые с флуоресцентным белком CFP (например, eCFP) или флуоресцентным белком YFP (например, eYFP) - тау4RD-CFP/тау4RD-YFP (TCY), где содержащий повторы домен тау-белка (4RD) содержит патогенную мутацию P301S. В данных биосенсорных линиях агрегация белков тау-CFP/тау-YFP производит сигнал резонансного переноса энергии флуоресценции (FRET) - результат переноса энергии флуоресценции от донора CFP к акцептору YFP. Термин CFP (голубой флуоресцентный белок) при использовании в контексте данного документа включает в себя eCFP (усиленный голубой флуоресцентный белок), а термин YFP (желтый флуоресцентный белок) при использовании в контексте данного документа включает в себя eYFP (усиленный желтый флуоресцентный белок). Клетки, положительные в отношении FRET, содержащие агрегаты тау-белка, могут быть отсортированы и выделены с помощью метода проточной цитометрии. В исходном состоянии нестимулированные клетки экспрессируют репортеры в стабильном растворимом виде с минимальным сигналом FRET. При стимуляции (например, липосомная трансфекция засеваемыми частицами) репортерные белки образуют агрегаты, производя сигнал FRET. Клетки, содержащие агрегаты, можно выделить с помощью метода FACS. Стабильно размножающиеся клеточные линии, содержащие агрегаты, Аг[+], могут быть выделены путем клонального серийного разведения клеточных линий Аг[-].
[00124] В данную линию клеток-биосенсоров тау-белка было внесено несколько модификаций, чтобы сделать ее пригодной для генетического скрининга с использованием библиотек CRISPRn. Сначала данные клетки-биосенсоры тау-белка были модифицированы путем введения Cas-экспрессирующего трансгена (например, Cas9 или SpCas9) для применения в скринингах CRISPRn. Затем были получены субклоны этих Cas-экспрессирующих линий-биосенсоров тау-белка, в которых тау-белок стабильно присутствует либо в неагрегированном (состояние по умолчанию) (Аг[-]), либо в агрегированном состоянии (Аг[+]).
[00125] Данные клеточные линии были использованы для разработки способа скрининга, в котором Cas-экспрессирующие клетки-биосенсоры тау-белка, либо содержащие агрегаты (Аг[+]), либо не содержащие агрегаты (Аг[-]), трансдуцировали с помощью библиотеки гидовых РНК CRISPR для введения нокаут-мутаций в каждый целевой ген. Затем был проведен скрининг с целью идентификации не только основных генов, состоящих из тех генов, чьи нацеленные егРНК со временем истощаются в линиях клеток Аг[+] и Аг[-], но и синтетические летальных генов, состоящих из генов, чьи нацеленные егРНК со временем истощаются преимущественно в линиях клеток Аг[+] по сравнению с линиями клеток Аг[-]. Профили истощения оценивались с использованием недавно определенного анализа динамики изменений во времени, в котором гидовые РНК с паттерном последовательного снижения считываний от самого раннего момента времени до конечного момента времени считались истощенными. Это новый аналитический подход к оценке истощения гидовой РНК по сравнению с более традиционным подходом, заключающимся в простом сравнении считываний коллекции конечных клеток с первым пассажем.
[00126] Аналогичным образом, в данную линию клеток-биосенсоров тау-белка было внесено несколько модификаций, чтобы сделать ее пригодной для генетического скрининга с использованием библиотек CRISPRa (например, для применения с системой медиатора синергической активации (SAM) CRISPR/Cas). В иллюстративной системе SAM несколько доменов активации взаимодействуют и вызывают активацию транскрипции, превышающую ту, которая могла бы быть вызвана любым одним фактором в одиночку. Например, иллюстративная система SAM включает в себя химерный белок Cas, который содержит белок Cas с инактивированной нуклеазной активностью, слитый с одним или большим числом доменов активации транскрипции (например, VP64), и химерный адаптерный белок, который содержит адаптерный белок (например, белок оболочки MS2 (МСР)), слитый с одним или большим числом доменов активации транскрипции (например, слитый с р65 и HSF1). МСР естественным образом связывается со шпильками MS2. В иллюстративной системе SAM белок МСР взаимодействует со стержневыми петлями MS2, сконструированными в CRISPR-ассоциированную егРНК, и тем самым перемещает связанные факторы транскрипции в соответствующее местоположение в геноме.
[00127] Сначала данные клетки-биосенсоры тау-белка были модифицированы путем введения одного или большего числа трансгенов, экспрессирующих химерный белок Cas, который содержит белок Cas с инактивированной нуклеазной активностью, слитый с одним или большим числом доменов активации транскрипции (например, VP64), и химерный адаптерный белок, который содержит адаптерный белок (например, белок оболочки MS2 (МСР)), слитый с одним или большим числом доменов активации транскрипции (например, слитый с р65 и HSF1). Несмотря на то, что в данном документе описаны системы SAM, также можно применять другие системы CRISPRa, такие как белок Cas с инактивированной нуклеазной активностью, слитый с одним или большим числом доменов активации транскрипции, при этом такие системы не включают в себя также и химерный адаптерный белок. В таких случаях клетки-биосенсоры тау-белка могут быть модифицированы путем введения трансгена, экспрессирующего химерный белок Cas.
[00128] Затем были получены субклоны этих Cas-экспрессирующих (т.е. экспрессирующих химерный Cas и экспрессирующих химерный адаптер) линий-биосенсоров тау-белка, в которых тау-белок стабильно присутствует либо в неагрегированном (состояние по умолчанию) (Аг[-]), либо в агрегированном состоянии (Аг[+]).
[00129] Данные клеточные линии были использованы для разработки способа скрининга, в котором SAM-экспрессирующие клетки-биосенсоры тау-белка, либо содержащие агрегаты (Аг[+]), либо не содержащие агрегаты (Аг[-]), трансдуцировали с помощью библиотеки гидовых РНК CRISPRa для активации транскрипции каждого целевого гена. Затем был проведен скрининг для выявления гена, который при активации имеет синтетический летальный эффект. В частности, егРНК, которые специфически истощены в клеточной линии Аг[+], но при этом не истощены или истощены в меньшей степени в клеточной линии Аг[-], могут указывать на синтетический летальный эффект, при котором активация конкретного целевого гена сочетается с наличием агрегатов тау-белка в клетке, чтобы вызвать гибель данной клетки. Профили истощения оценивались с использованием недавно определенного анализа динамики изменений во времени, в котором гидовые РНК с паттерном последовательного снижения считываний от самого раннего момента времени до конечного момента времени считались истощенными. Это новый аналитический подход к оценке истощения гидовой РНК по сравнению с более традиционным подходом, заключающимся в простом сравнении считываний коллекции конечных клеток с первым пассажем. Эти синтетические летальные гены представляют интерес как потенциальные модификаторы ассоциированной с тау-белком клеточной токсичности.
II. Линии биосенсорных клеток Cas/may и SAM/may, и способы их получения
А. Биосенсорные клетки Cas/тау и биосенсорные клетки SAM/тау
[00130] В данном документе описаны клетки, не только экспрессирующие первый содержащий повторы домен тау-белка (например, содержащий домен связывания микротрубочек (MBD) тау-белка), связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, но также экспрессирующие белок Cas, такой как Cas9. В данном документе также описаны клетки, не только экспрессирующие первый содержащий повторы домен тау-белка (например, содержащий домен связывания микротрубочек (MBD) тау-белка), связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, но также экспрессирующие химерный белок Cas, который содержит белок Cas с инактивированной нуклеазной активностью, слитый с одним или большим числом доменов активации транскрипции, и химерный адаптерный белок, который содержит адаптерный белок, слитый с одним или большим числом доменов активации транскрипции. Первый содержащий повторы домен тау-белка, связанный с первым репортером, может стабильно экспрессироваться, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, может стабильно экспрессироваться. Например, ДНК, кодирующая первый содержащий повторы домен тау-белка, связанный с первым репортером, может быть геномно интегрирована, и ДНК, кодирующая второй содержащий повторы домен тау-белка, связанный со вторым репортером, может быть геномно интегрирована. Подобным образом, белок Cas может стабильно экспрессироваться в биосенсорных клетках Cas/тау. Например, ДНК, кодирующая белок Cas, может быть геномно интегрирована. Аналогичным образом, химерный белок Cas и (или) химерный адаптерный белок могут стабильно экспрессироваться в биосенсорных клетках SAM/тау. Например, ДНК, кодирующая химерный белок Cas, может быть геномно интегрирована, и (или) ДНК, кодирующая химерный адаптерный белок, может быть геномно интегрирована.
1. Тау-белок и содержащий повторы домен тау-белка, связанный с репортерами
[00131] Связанный с микротрубочками тау-белок представляет собой белок, который способствует сборке и стабильности микротрубочек, и преимущественно экспрессируется в нейронах. Тау-белок играет роль в стабилизации микротрубочек нейронов и, таким образом, способствует разрастанию аксонов. При болезни Альцгеймера (БА) и семействе родственных нейродегенеративных заболеваний, называемых таупатиями, тау-белок аномально гиперфосфорилирован и агрегирован в пучки филаментов (парные спиральные нити), которые проявляются в виде нейрофибриллярных клубков. Таупатии представляют собой группу гетерогенных нейродегенеративных патологических состояний, характеризующихся отложением аномального тау-белка в головном мозге.
[00132] Содержащий повторы домен тау-белка может происходить из тау-белка любого животного или млекопитающего, такого как человек, мышь или крыса. В одном конкретном примере содержащий повторы домен тау-белка происходит из тау-белка человека. Иллюстративному тау-белку человека присвоен учетный номер UniProt Р10636. Тау-белки являются продуктами альтернативного сплайсинга из одного гена, который у человека обозначается как МАРТ (связанный с микротрубочками тау-белок). Содержащий повторы домен тау-белка несет в себе мотивы последовательности, ответственные за агрегацию (т.е. это домен из тау-белка, склонный к агрегации). В зависимости от сплайсинга содержащий повторы домен тау-белка содержит либо три, либо четыре повторяющихся области, которые составляют склонное к агрегации ядро данного белка, которое часто называют содержащим повторы доменом (RD). В частности, содержащий повторы домен тау-белка представляет собой ядро области связывания микротрубочек и несет в себе гексапептидные мотивы в R2 и R3, которые ответственны за агрегацию тау-белка. В головном мозге человека существует шесть изоформ тау-белка, длина которых варьирует от 352 до 441 аминокислоты. Данные изоформы различаются на карбоксильном конце в зависимости от наличия трех или четырех содержащих повторы доменов (R1-R4) в дополнение к наличию или отсутствию одного или двух содержащих вставки доменов на аминоконце. Содержащие повторы домены, расположенные в карбоксиконцевой половине тау-белка, как полагают, важны для связывания микротрубочек, а также для патологической агрегации тау-белка в парные спиральные филаменты (ПСФ, англ. «PHF»), которые являются основными составляющими нейрофибриллярных клубков, обнаруживаемых при таупатиях. Иллюстративные последовательности для четырех содержащих повторы доменов (R1-R4) представлены в SEQ ID NO: 1-4, соответственно. Иллюстративные кодирующие последовательности для четырех содержащих повторы доменов (R1-R4) представлены в SEQ ID NO: 5-8. Иллюстративная последовательность для домена тау-белка с четырьмя повторами представлена в SEQ ID NO: 9. Иллюстративная кодирующая последовательность для домена тау-белка с четырьмя повторами представлена в SEQ ID NO: 10. Иллюстративная последовательность для домена тау-белка с четырьмя повторами с мутацией P301S представлена в SEQ ID NO: 11. Иллюстративная кодирующая последовательность для домена тау-белка с четырьмя повторами с мутацией P301S представлена в SEQ ID NO: 12.
[00133] Содержащий повторы домен тау-белка, используемый в биосенсорных клетках Cas/тау или биосенсорных клетках SAM/тау, может содержать связывающий микротрубочки домен тау-белка (MBD). Содержащий повторы домен тау-белка, используемый в биосенсорных клетках Cas/тау или биосенсорных клетках SAM/тау, может содержать один, или большее число, или все четыре содержащих повторы домена (R1-R4). Например, содержащий повторы домен тау-белка может содержать, состоять по существу из, или состоять из одной, или большего числа, или всех из последовательностей с SEQ ID NO: 1, 2, 3 и 4, или из последовательностей, которые по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентичны последовательностям с SEQ ID NO: 1, 2, 3 и 4. В одном конкретном примере содержащий повторы домен тау-белка представляет собой домен тау-белка с четырьмя повторами (R1-R4), обнаруживаемый в нескольких изоформах тау-белка. Например, содержащий повторы домен тау-белка может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 9 или последовательности с SEQ ID NO: 11, или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 9 или последовательности с SEQ ID NO: 11. В одном конкретном примере нуклеиновая кислота, кодирующая содержащий повторы домен тау-белка, может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 12 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 12, необязательно при этом данная нуклеиновая кислота кодирует белок, содержащий, состоящий по существу из, или состоящий из последовательности с SEQ ID NO: 11. В другом конкретном примере нуклеиновая кислота, кодирующая второй содержащий повторы домен тау-белка, связанный со вторым репортером, может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 10 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 10, необязательно при этом данная нуклеиновая кислота кодирует белок, содержащий, состоящий по существу из, или состоящий из последовательности с SEQ ID NO: 9. Первый и второй содержащие повторы домены тау-белка в клетках, описанных в данном документе, могут быть одинаковыми, похожими или разными.
[00134] Один или оба из указанных первого содержащего повторы домена тау-белка, связанного с первым репортером, и второго содержащего повторы домена тау-белка, связанного со вторым репортером, могут стабильно экспрессироваться в клетках. Например, нуклеиновые кислоты, кодирующие один или оба из указанных первого содержащего повторы домена тау-белка, связанного с первым репортером, и второго содержащего повторы домена тау-белка, связанного со вторым репортером, могут быть геномно интегрированы в популяцию клеток и функционально связаны с промоторами, активными в клетке.
[00135] Содержащие повторы домены тау-белка, используемые в клетках, описанных в данном документе, также могут содержать патогенную мутацию тау-белка, такую как мутация, способствующая агрегации. Такая мутация может представлять собой, например, мутацию, которая связана с (например, сегрегируется с) таупатией или вызывает таупатию. В качестве одного из примеров, указанная мутация может представлять собой сенсибилизирующую к агрегации мутацию, которая сенсибилизирует тау-белок к засеву, но не приводит к тому, что тау-белок легко агрегирует сам по себе. Например, указанная мутация может представлять собой мутацию P301S, связанную с заболеванием. Под мутацией P301S понимается мутация P301S тау-белка человека или соответствующая мутация в другом тау-белке при оптимальном выравнивании с тау-белком человека. Мутация P301S в тау-белке проявляет высокую сенсибилизацию к засеву, но не приводит к агрегации сама по себе. Таким образом, хотя на исходном уровне репортерные тау-белки, содержащие мутацию P301S, существуют в клетке в стабильной растворимой форме, воздействие экзогенных факторов засева тау-белка приводит к агрегации репортерного тау-белка. Другие мутации тау-белка включают в себя, например, K280del, P301L, V337M, P301L/V337M и K280del/I227P/I308P.
[00136] Первый содержащий повторы домен тау-белка может быть связан с первым репортером, а второй содержащий повторы домен тау-белка может быть связан со вторым репортером любыми способами. Например, репортер может быть слит с содержащим повторы доменом тау-белка (например, как часть слитого белка).
[00137] Первый репортер и второй репортер могут представлять собой пару уникальных репортеров, которые могут действовать вместе как внутриклеточный биосенсор, который производит детектируемый сигнал при агрегировании первого и второго белков. В качестве одного из примеров, репортеры могут представлять собой флуоресцентные белки, и для измерения агрегации белков может быть использован резонансный перенос энергии флуоресценции (FRET). В частности, первый и второй репортеры могут представлять собой FRET-пару. Примеры FRET-nap (донорный и акцепторный флуорофоры) хорошо известны. См., например, работу Bajar et al. (2016) Sensors (Basel) 16 (9): 1488, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей. Иллюстративные методики флуоресцентной микроскопии основаны на поглощении флуорофором света при волне одной длины (возбуждение) с последующим испусканием вторичной флуоресценции при волне большей длины. Механизм резонансной передачи энергии флуоресценции включает в себя донорный флуорофор в возбужденном электронном состоянии, который может передавать свою энергию возбуждения соседнему акцепторному хромофору безызлучательным образом посредством дальнодействующих диполь-дипольных взаимодействий. Например, донор энергии FRET может представлять собой первый репортер, а акцептор энергии FRET может представлять собой второй репортер. В качестве альтернативы, донор энергии FRET может представлять собой второй репортер, а акцептор энергии FRET может представлять собой первый репортер. В конкретном примере первый и второй репортеры представляют собой CFP и YFP. Иллюстративные белковые и кодирующие последовательности для CFP представлены, например, в SEQ ID NO: 13 и 14, соответственно. Иллюстративные белковые и кодирующие последовательности для YFP представлены, например, в SEQ ID NO: 15 и 16, соответственно. В качестве конкретного примера, CFP может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 13 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 13. В качестве другого конкретного примера, YFP может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 15 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 15.
[00138] В качестве другого примера можно использовать стратегию комплементации белковых фрагментов для обнаружения агрегации. Например, сплит-люцифераза может использоваться для получения биолюминесценции из субстрата, а первый и второй репортеры могут представлять собой амино- (NLuc) и карбокси- (CLuc) концевые фрагменты люциферазы. Примеры люциферазы включают в себя люциферазу Renilla, люциферазу светлячка, люциферазу жука-щелкуна и люциферазу Metridia.
[00139] В одном неограничивающем примере биосенсорные клетки, описанные в данном документе, содержат два трансгена, стабильно экспрессирующих варианты ассоциированного с заболеванием тау-белка, слитые с флуоресцентным белком CFP или флуоресцентным белком YFP, соответственно (тау4RD-CFP/тау4RD-YFP (TCY)), при этом содержащий четыре повтора домен тау-белка (4RD) содержит патогенную мутацию P301S. В данных биосенсорных линиях агрегация белков тау-CFP/тау-YFP производит FRET-сигнал, который является результатом переноса энергии флуоресценции от донора CFP к акцептору YFP. Клетки, положительные в отношении FRET, содержащие агрегаты тау-белка, могут быть отсортированы и выделены с помощью метода проточной цитометрии. В исходном состоянии нестимулированные клетки экспрессируют репортеры в стабильном растворимом виде с минимальным сигналом FRET. При стимуляции (например, липосомная трансфекция засеваемыми частицами) репортерные белки образуют агрегаты, производя сигнал FRET.
[00140] Описанные в данном документе биосенсорные клетки Cas/тау могут быть положительными в отношении агрегации тау-белка (Аг[+]) клетками, в которых содержащий повторы домен тау-белка стабильно присутствует в агрегированном состоянии, что означает, что агрегаты содержащего повторы домена тау-белка стабильно сохраняются во всех клетках при росте и множественных пассажах со временем. В качестве альтернативы, описанные в данном документе биосенсорные клетки Cas/тау могут быть отрицательными в отношении агрегации тау-белка (Аг[-]).
2. Белки Cas и химерные белки Cos
[00141] Описанные в данном документе биосенсорные клетки Cas/тау также содержат нуклеиновые кислоты (ДНК или РНК), кодирующие белки Cas. Необязательно, указанный белок Cas стабильно экспрессируется. Необязательно, указанные клетки содержат геномно интегрированную последовательность, кодирующую белок Cas. Аналогичным образом, биосенсорные клетки SAM/тау, описанные в данном документе, также содержат нуклеиновые кислоты (ДНК или РНК), кодирующие химерные белки Cas, содержащие белок Cas с инактивированной нуклеазной активностью, слитый с одним или большим числом доменов активации транскрипции (например, VP64). Необязательно, указанный химерный белок Cas стабильно экспрессируется. Необязательно, указанные клетки содержат геномно интегрированную последовательность, кодирующую химерный белок Cas.
[00142] Белки Cas являются частью систем коротких палиндромных повторов, регулярно расположенных группами (CRISPR)/связанных с CRISPR (Cas) систем. Системы CRISPR/Cas включают в себя транскрипты и другие элементы, участвующие в экспрессии генов Cas или регулирующие активность генов Cas. Система CRISPR/Cas может представлять собой, например, систему типа I, систему типа II, систему типа III или систему типа V (например, подтип VA или подтип VB). В описанных в данном документе способах и композициях можно применять системы CRISPR/Cas путем использования комплексов CRISPR (содержащих гидовую РНК (гРНК) в комплексе с белком Cas) для сайт-направленного связывания или расщепления нуклеиновых кислот.
[00143] Системы CRISPR/Cas, применяемые в описанных в данном документе композициях и способах, могут быть не встречающимися в природе. «Не встречающаяся в природе» система включает в себя то, что было создано деятельностью человека, например, один или большее число компонентов системы, измененных или мутированных по сравнению с тем состоянием, в котором они встречаются в природе, по меньшей мере по существу не содержащих по меньшей мере одного другого компонента, с которым они естественным образом связаны в природе, или связанных по меньшей мере с одним другим компонентом, с которым они не связаны естественным образом. Например, в некоторых системах CRISPR/Cas применяются не встречающиеся в природе комплексы CRISPR, содержащие гРНК и белок Cas, которые не встречаются вместе в природе, применяется белок Cas, который не встречается в природе, или применяется иРНК, которая не встречается в природе.
[00144] Белки Cas обычно содержат по меньшей мере один домен, распознающий или связывающий РНК, который может взаимодействовать с гидовыми РНК. Белки Cas также могут содержать нуклеазные домены (например, ДНКазные домены или РНКазные домены), ДНК-связывающие домены, геликазные домены, домены межбелкового взаимодействия, домены димеризации и другие домены. Некоторые такие домены (например, домены ДНКазы) могут происходить из природного белка Cas. Другие такие домены могут быть добавлены для получения модифицированного белка Cas. Нуклеазный домен обладает каталитической активностью в отношении расщепления нуклеиновой кислоты, которое включает в себя разрыв ковалентных связей молекулы нуклеиновой кислоты. Расщепление может приводить к образованию тупых или ступенчатых концов, и оно может быть одно цепочечным или двухцепочечным. Например, белок Cas9 дикого типа обычно создает продукт расщепления с тупыми концами. В качестве альтернативы, белок Cpf1 дикого типа (например, FnCpf1) может приводить к образованию продукта расщепления с 5'-выступом из 5 нуклеотидов, при этом расщепление происходит после 18-й пары оснований из последовательности РАМ в нецелевой цепи и после 23-го основания в целевой цепи. Белок Cas может обладать полной активностью расщепления для создания двухцепочечного разрыва в целевом геномном локусе (например, двухцепочечного разрыва с тупыми концами), или может представлять собой никазу, которая создает одноцепочечный разрыв в целевом геномном локусе.
[00145] Примеры белков Cas включают в себя Cas1, Cas1B, Cas2, Cas3, Cas4, Cas5, Cas5e (CasD), Cas6, Cas6e, Cas6f, Cas7, Cas8a1, Cas8a2, Cas8b, Cas8c, Cas9 (Csn1 или Csx12), Cas10, Cas10d, CasF, CasG, CasH, Csyl, Csy2, Csy3, Csel (CasA), Cse2 (CasB), Cse3 (CasE), Cse4 (CasC), Cscl, Csc2, Csa5, Csn2, Csm2, Csm3, Csm4, Csm5, Csm6, Cmr1, Cmr3, Cmr4, Cmr5, Cmr6, Csb1, Csb2, Csb3, Csx17, Csx14, Csx10, Csx16, CsaX, Csx3, Csx1, Csx15, Csf1, Csf2, Csi3, Csf4 и Cul966, и их модифицированные версии и их гомологи.
[00146] Иллюстративный белок Cas представляет собой белок Cas9 или белок, полученный из белка Cas9. Белки Cas9 происходят из системы CRISPR/Cas типа II и обычно имеют четыре ключевых мотива с консервативной архитектурой. Мотивы 1, 2 и 4 представляют собой мотивы, подобные RuvC, а мотив 3 представляет собой мотив HNH. Иллюстративные белки Cas9 происходят из Streptococcus pyogenes, Streptococcus thermophilus, Streptococcus sp., Staphylococcus aureus, Nocardiopsis dassonvillei, Streptomyces pristinaespiralis, Streptomyces viridochromogenes, Streptomyces viridochromogenes, Streptosporangium roseum, Streptosporangium roseum, Alicyclobacillus acidocaldarius, Bacillus pseudomycoides, Bacillus selenitireducens, Exiguobacterium sibiricum, Lactobacillus delbrueckii, Lactobacillus salivarius, Microscilla marina, Burkholderiales bacterium, Polaromonas naphthalenivorans, Polaromonas sp., Crocosphaera watsonii, Cyanothece sp., Microcystis aeruginosa, Synechococcus sp., Acetohalobium arabaticum, Ammonifex degensii, Caldicelulosiruptor becscii, Candidatus Desulforudis, Clostridium botulinum, Clostridium difficile, Finegoldia magna, Natranaerobius thermophilus, Pelotomaculum thermopropionicum, Acidithiobacillus caldus, Acidithiobacillus ferrooxidans, Allochromatium vinosum, Marinobacter sp., Nitrosococcus halophilus, Nitrosococcus watsoni, Pseudoalteromonas haloplanktis, Ktedonobacter racemifer, Methanohalobium evestigatum, Anabaena variabilis, Nodularia spumigena, Nostoc sp., Arthrospira maxima, Arthrospiraplatensis, Arthrospira sp., Lyngbya sp., Microcoleus chthonoplastes, Oscillatoria sp., Petrotoga mobilis, Thermosipho africanus, Acaryochloris marina, Neisseria meningitidis или Campylobacter jejuni. Дополнительные примеры представителей семейства Cas9 описаны в патентном документе WO 2014/131833, который включен в данный документ посредством ссылки во всей своей полноте для всех целей. Cas9 из S. pyogenes (SpCas9) (которому присвоен учетный номер SwissProt Q99ZW2) представляет собой иллюстративный белок Cas9. Иллюстративный белок SpCas9 и его кодирующая последовательность указаны в SEQ ID NO: 21 и SEQ ID NO: 22, соответственно. Cas9 из S. aureus (SaCas9) (которому присвоен учетный номер UniProt J7RUA5) представляет собой еще один иллюстративный белок Cas9. Cas9 из Campylobacter jejuni (CjCas9) (которому присвоен учетный номер UniProt Q0P897) представляет собой еще один иллюстративный белок Cas9. См., например, работу Kim et al. (2017) Nat. Comm. 8: 14500, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей. SaCas9 имеет меньший размер, чем SpCas9, а CjCas9 имеет меньший размер, чем оба белка SaCas9 и SpCas9. Cas9 из Neisseria meningitidis (Nme2Cas9) представляет собой еще один иллюстративный белок Cas9. См., например, работу Edraki et al. (2019) Mol. Cell 73 (4): 714-726, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей. Белки Cas9 из Streptococcus thermophilus (например, белок Cas9 из Streptococcus thermophilus LMD-9, кодируемый локусом CRISPR1 (StlCas9), или белок Cas9 из Streptococcus thermophilus, кодируемый локусом CRISPR3 (St3Cas9)) представляют собой другие иллюстративные белки Cas9. Белок Cas9 из Francisella novicida (FnCas9) или вариантный белок Cas9 RHA из Francisella novicida, который распознает альтернативный РАМ (замены E1369R/E1449H/R1556A) представляют собой другие иллюстративные белки Cas9. Обзор этих и других иллюстративных белков Cas9 представлен, например, в работе Cebrian-Serrano andDavies (2017) Маmm. Genome 28 (7): 247-261, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей.
[00147] В качестве одного из примеров, белок Cas может представлять собой белок Cas9. Например, белок Cas9 представлять собой белок Cas9 из Streptococcus pyogenes. В качестве одного конкретного примера, белок Cas может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 21 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 21. В качестве другого конкретного примера, химерный белок Cas, содержащий белок Cas с инактивированной нуклеазной активностью и один или большее число доменов активации транскрипции, может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 36 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 36.
[00148] Еще один иллюстративный белок Cas представляет собой белок Cpfl (CRISPR из Prevotella и Francisella 1). Cpf1 представляет собой большой белок (около 1300 аминокислот), который содержит RuvC-подобный нуклеазный домен, гомологичный соответствующему домену Cas9, вместе с аналогом характерного богатого аргинином кластера из Cas9. Тем не менее, в белке Cpf1 отсутствует нуклеазный домен HNH, который присутствует в белках Cas9, а RuvC-подобный домен является смежным в последовательности Cpfl, в отличие от Cas9, который содержит длинные вставки, включая домен HNH. См., например, работу Zetsche et al. (2015) Cell 163 (3): 759-771, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей. Иллюстративные белки Cpf1 происходят из Francisella tularensis 1, Francisella tularensis subsp.novicida, Prevotella albensis, Lachnospiraceae bacterium MC2017 1, Butyrivibrio proteoclasticus, Peregrinibacteria bacterium GW2011 GWA2 33 10, Parcubacteria bacterium GW2011GWC2 44 17, Smithella sp.SCADC, Acidaminococcus sp.BV3L6, Lachnospiraceae bacterium MA2020, Candidatus Methanoplasma termitum, Eubacterium eligens, Moraxella bovoculi 237, Leptospira inadai, Lachnospiraceae bacterium ND2006, Porphyromonas crevioricanis 3, Prevotella disiens и Porphyromonas macacae. Cpfl из Francisella novicida U112 (FnCpf1; присвоен учетный номер UniProt A0Q7Q2) представляет собой иллюстративный белок Cpf1.
[00149] Белки Cas могут представлять собой белки дикого типа (т.е. белки, которые встречаются в природе), модифицированные белки Cas (т.е. вариантные белки Cas) или фрагменты белков Cas дикого типа, или модифицированные белки Cas. Белки Cas также могут представлять собой варианты или фрагменты, активные в отношении каталитической активности белков Cas дикого типа или модифицированных белков Cas. Варианты или фрагменты, активные в отношении каталитической активности, могут быть по меньшей мере на 80%, на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или более идентичны последовательности белка Cas дикого типа или последовательности модифицированного белка Cas, или его части, при этом активные варианты сохраняют способность разрезать в желаемом сайте расщепления и, следовательно, сохраняют активность, индуцирующую двухцепочечный разрыв. Анализы на предмет активности, индуцирующей двухцепочечный разрыв, известны и обычно измеряют общую активность и специфичность белка Cas на субстратах ДНК, содержащих сайт расщепления.
[00150] Одним из примеров модифицированного белка Cas является модифицированный белок SpCas9-HF1, который представляет собой высокоточный вариант Cas9 из Streptococcus pyogenes, несущий изменения (N497A/R661A/Q695A/Q926A), предназначенные для снижения неспецифических контактов с ДНК. См., например, работу Kleinstiver et al. (2016) Nature 529 (7587): 490-495, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей. Другим примером модифицированного белка Cas является модифицированный вариант eSpCas9 (K848A/K1003A/R1060A), разработанный для снижения нецелевых эффектов. См., например, работу Slaymaker et al. (2016) Science 351 (6268): 84-88, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей. Другие варианты SpCas9 включают в себя К855А и K810A/K1003A/R1060A. Обзор этих и других модифицированных белков Cas представлен, например, в работе Cebrian-Serrano and Davies (2017) Матт.Genome 28 (7): 247-261, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей. Другим примером модифицированного белка Cas9 является xCas9, который представляет собой вариант SpCas9, который может распознавать более широкий диапазон последовательностей РАМ. См., например, работу Ни et al. (2018)Nature 556: 57-63, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей.
[00151] Белки Cas могут быть модифицированы с целью увеличения или уменьшения одного или большего числа из следующих параметров: аффинность связывания нуклеиновой кислоты, специфичность связывания нуклеиновой кислоты и ферментативная активность. Белки Cas также могут быть модифицированы с целью изменения любой другой активности или свойства белка, например, стабильности. Например, белок Cas может быть усечен для удаления доменов, которые не являются существенными для функции белка, или для оптимизации (например, повышения или понижения) активности или свойства белка Cas. В качестве другого примера, один или большее число нуклеазных доменов белка Cas могут быть модифицированы, удалены или инактивированы (например, для использования в биосенсорных клетках SAM/тау, содержащих белок Cas с инактивированной нуклеазной активностью).
[00152] Белки Cas могут содержать по меньшей мере один нуклеазный домен, такой как ДНКазный домен. Например, белок Cpf1 дикого типа обычно содержит RuvC-подобный домен, который расщепляет обе цепи целевой ДНК, возможно, в димерной конфигурации. Белки Cas также могут содержать по меньшей мере два нуклеазных домена, таких как ДНКазные домены. Например, белок Cas9 дикого типа обычно содержит RuvC-подобный нуклеазный домен и HNH-подобный нуклеазный домен. Каждый из доменов RuvC и HNH может разрезать разные цепи двухцепочечной ДНК с образованием двухцепочечного разрыва в ДНК. См., например, работу Jinek et al. (2012) Science 337: 816-821, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей.
[00153] Один, или большее число, или все нуклеазные домены могут быть удалены или мутированы таким образом, что они больше не функциональны или имеют пониженную нуклеазную активность. Например, если один из нуклеазных доменов в белке Cas9 удален или мутирован, то полученный в результате белок Cas9 может называться никазой и может генерировать одноцепочечный разрыв в двухцепочечной целевой ДНК, но не двухцепочечный разрыв (т.е. он может расщеплять комплементарную цепь или некомплементарную цепь, но не обе). Если оба нуклеазных домена удалены или мутированы, то полученный в результате белок Cas (например, Cas9) будет иметь пониженную способность расщеплять обе цепи двухцепочечной ДНК (например, белок Cas с нулевой нуклеазной активностью или белок Cas с инактивированной нуклеазной активностью, или каталитически мертвый белок Cas (dCas)). Примером мутации, которая превращает Cas9 в никазу, является мутация D10A (аспартат на аланин в положении 10 белка Cas9) в домене RuvC белка Cas9 из S. pyogenes. Аналогичным образом, Н939А (гистидин на аланин в положении аминокислоты 839), Н840А (гистидин на аланин в положении аминокислоты 840) или N863A (аспарагин на аланин в положении аминокислоты N863) в домене HNH белка Cas9 из S. pyogenes может превращать Cas9 в никазу. Другие примеры мутаций, которые превращают Cas9 в никазу, включают в себя соответствующие мутации белка Cas9 из S. thermophilus. См., например, Sapranauskas et al. (2011) Nucleic Acids Res. 39 (21): 9275 - 9282 и патентный документ WO 2013/141680, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. Такие мутации могут быть получены с использованием таких способов, как сайт-направленный мутагенез, ГТЦР-опосредованный мутагенез или полный генный синтез. Примеры других мутаций, создающих никазы, можно найти, например, в патентных документах WO 2013/176772 и WO 2013/142578, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. Если в белке Cas удалены или мутированы все нуклеазные домены (например, в белке Cas9 удалены или мутированы оба нуклеазных домена), то полученный в результате белок Cas (например, Cas9) будет иметь пониженную способность расщеплять обе цепи двухцепочечной ДНК (например, белок Cas с нулевой нуклеазной активностью или с белок Cas инактивированной нуклеазной активностью). Одним из конкретных примеров является двойной мутант D10A/H840A белка Cas9 из S. pyogenes или соответствующий двойной мутант белка Cas9 из другого вида при оптимальном выравнивании с последовательностью Cas9 из S. pyogenes. Другим конкретным примером является двойной мутант D10A/N863A белка Cas9 из S. pyogenes или соответствующий двойной мутант белка Cas9 из другого вида при оптимальном выравнивании с последовательностью Cas9 из S. pyogenes.
[00154] Примеры инактивирующих мутаций в каталитических доменах xCas9 являются такими же, как описанные выше примеры для SpCas9. Также известны примеры инактивирующих мутаций в каталитических доменах белков Cas9 из Staphylococcus aureus. Например, фермент Cas9 из Staphylococcus aureus (SaCas9) может включать в себя замену в положении N580 (например, замену N580A) и замену в положении D10 (например, замену D10A) для образования белка Cas с инактивированной нуклеазной активностью. См., например, патентный документ WO 2016/106236, который включен в данный документ посредством ссылки во всей своей полноте для всех целей. Также известны примеры инактивирующих мутаций в каталитических доменах Nme2Cas9 (например, комбинация D16A и Н588А). Также известны примеры инактивирующих мутаций в каталитических доменах StlCas9 (например, комбинация D9A, D598A, Н599А и N622A). Также известны примеры инактивирующих мутаций в каталитических доменах St3Cas9 (например, комбинация D10A и N870A). Также известны примеры инактивирующих мутаций в каталитических доменах CjCas9 (например, комбинация D8A и Н559А). Также известны примеры инактивирующих мутаций в каталитических доменах FnCas9 и RHA FnCas9 (например, N995A).
[00155] Также известны примеры инактивирующих мутаций в каталитических доменах белков Cpfl. Что касается белков Cpfl из Francisella novicida U112 (FnCpfl), Acidaminococcus sp.BV3L6 (AsCpfl), Lachnospiraceae bacterium ND2006 (LbCpfl) и Moraxella bovoculi 237 (MbCpfl Cpfl), такие мутации могут включать в себя мутации в положениях 908, 993 или 1263 в AsCpfl, или в соответствующих положениях в ортологах Cpfl, либо в положениях 832, 925, 947 или 1180 в LbCpfl, или в соответствующих положениях в ортологах Cpfl. Такие мутации могут включать в себя, например, одну или большее число из мутаций D908A, Е993А и D1263A в AsCpfl, или соответствующие мутации в ортологах Cpfl, либо D832A, Е925А, D947A и D1180A в LbCpfl, или соответствующие мутации в ортологах Cpfl. См., например, патентный документ US 2016/0208243, который включен в данный документ посредством ссылки во всей своей полноте для всех целей.
[00156] Белки Cas также могут быть функционально связаны с гетерологичными полипептидами в качестве слитых белков. Например, белок Cas может быть слит с доменом расщепления, доменом эпигенетической модификации, доменом активации транскрипции или доменом репрессии транскрипции. См. патентный документ WO 2014/089290, который включен в данный документ посредством ссылки во всей своей полноте для всех целей. Например, белки Cas могут быть функционально связаны или слиты с доменом активации транскрипции для применения в биосенсорных клетках SAM/тау. Примеры доменов активации транскрипции включают в себя домен активации VP16 вируса простого герпеса, VP64 (который является тетрамерным производным от VP16), домен активации р65 NFκB, домены активации р53 1 и 2, домен активации CREB (белка, связывающего элемент отклика цАМФ), домен активации Е2А и домен активации NFAT (ядерного фактора активированных Т-клеток). Другие примеры включают в себя домены активации из Octl, Oct-2A, SP1, АР-2, CTF1, Р300, СВР, PCAF, SRC1, PvALF, ERF-2, OsGAI, HALF-1, CI, API, ARF-5, ARF-6, ARF-7, ARF-8, CPRF1, CPRF4, MYC-RP/GP, TRAB1PC4 и HSF1. См., например, US 2016/0237456, EP3045537 и 2011/146121, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. В некоторых случаях может применяться система активации транскрипции, содержащая слитый белок dCas9-VP64, спаренный с MS2-p65-HSF1. Гидовые РНК в таких системах могут быть сконструированы с последовательностями аптамера, присоединенными к тетрапетле егРНК и шпильке 2, предназначенным для связывания димеризованных белков оболочки бактериофага MS2. См., например, работу Konermann et al. (2015) Nature 517 (7536): 583 - 588, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей. Примеры доменов репрессии транскрипции включают в себя индуцируемые домены раннего репрессора цАМФ (ICER), репрессорные домены Kruppel-ассоциированного бокса A (KRAB-А), богатые глицином репрессорные домены YY1, Spl-подобные репрессоры, E(spl) репрессоры, IκВ репрессор и МеСР2. Другие примеры включают в себя домены репрессии транскрипции из А/В, КОХ, ТФР-бета-индуцирумого раннего гена (TIEG), v-erbA, SID, SID4X, MBD2, MBD3, DNMT1, DNMG3A, DNMT3B, Rb, ROM2, см., например, ЕР3045537 и WO 2011/146121, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. Белки Cas также могут быть слиты с гетерологичным полипептидом, обеспечивающим повышенную или пониженную стабильность. Слитый домен или гетерологичный полипептид могут быть расположены на N-конце, С-конце или внутри белка Cas.
[00157] Белки Cas также могут быть функционально связаны с гетерологичными полипептидами в качестве слитых белков. В качестве одного из примеров, белок Cas может быть слит с одним или большим числом гетерологичных полипептидов, которые обеспечивают субклеточную локализацию. Такие гетерологичные полипептиды могут включать в себя, например, один или большее число из сигналов ядерной локализации (NLS), таких как монопартитный NLS SV40 и (или) бипартитный NLS импортина альфа для нацеливания на ядро, сигнал митохондриальной локализации для нацеливания на митохондрии, сигнал удержания в ЭР и т.п. См., например, работу Lange et al. (2007) J. Biol. Chem. 282: 5101-5105, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей. Такие сигналы субклеточной локализации могут быть расположены на N-конце, С-конце или в любой области в пределах белка Cas. NLS может содержать отрезок основных аминокислот и может быть монопартитной последовательностью или бипартитной последовательностью. Необязательно, белок Cas может содержать два или большее число NLS, включая NLS (например, NLS импортина альфа или монопартитный NLS) на N-конце и NLS (например, NLS SV40 или бипартитный NLS) на С-конце. Белок Cas также может содержать два или большее число NLS на N-конце и (или) два или большее число NLS на С-конце.
[00158] Белки Cas также могут быть функционально связаны с проникающим в клетку доменом, или с доменом трансдукции белка. Например, проникающий в клетку домен может происходить из белка ТАТ ВИЧ-1, проникающего в клетку мотива TLM из вируса гепатита В человека, MPG, Рер-1, VP22, проникающего в клетку белка из вируса простого герпеса, или из полиаргининовой пептидной последовательности. См., например, WO 2014/089290 и US 2013/176772, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. Проникающий в клетку домен может быть расположен на N-конце, С-конце или в любой области в пределах белка Cas.
[00159] Белки Cas также могут быть функционально связаны с гетерологичный полипептидом для облегчения отслеживания или очистки, например, с флуоресцентным белком, меткой очистки или меткой эпитопа. Примеры флуоресцентных белков включают в себя зеленые флуоресцентные белки (например, GFP, GFP-2, tagGFP, turboGFP, eGFP, Emerald, Azami Green, мономерный Azami Green, CopGFP, AceGFP, ZsGreenl), желтые флуоресцентные белки (например, YFP, eYFP, Citrine, Venus, YPet, PhiYFP, ZsYellowl), синие флуоресцентные белки (например, BFP, eBFP, eBFP2, Azurite, mKalamal, GFPuv, Sapphire, T-sapphire), голубые флуоресцентные белки (например, CFP, eCFP, Cerulean, CyPet, AmCyanl, Midoriishi-Cyan), красные флуоресцентные белки (например, RFP, mKate, mKate2, mPlum, мономер DsRed, mCherry, mRFPl, DsRed-Express, DsRed2, мономер DsRed, тандем HcRed, HcRedl, AsRed2, eqFP611, mRaspberry, mStrawberry, Jred), оранжевые флуоресцентные белки (например, mOrange, mKO, Kusabira-Orange, мономерный Kusabira-Orange, mTangerine, tdTomato) и любой другой подходящий флуоресцентный белок. Примеры меток включают в себя глутатион-S-трансферазу (GST), хитин-связывающий белок (СВР), мальтозо-связывающий белок, тиоредоксин (TRX), поли(NANP), метку тандемной аффинной очистки (ТАР), myc, AcV5, AU1, AU5, Е, ECS, Е2, FLAG, гемагглютинин (НА), nus, Softag 1, Softag 3, Strep, SBP, Glu-Glu, HSV, KT3, S, S1, T7, V5, VSV-G, гистидин (His), белок-носитель карбоксила биотина (BCCP) и кальмодулин.
[00160] Белки Cas могут быть предоставлены в любом виде. Например, белок Cas может быть предоставлен в виде белка. Например, белок Cas может быть представлен в виде белка Cas в комплексе с гРНК. В качестве альтернативы, белок Cas может быть предоставлен в виде нуклеиновой кислоты, кодирующей белок Cas, например, такой нуклеиновой кислоты, как РНК (например, информационная РНК (иРНК)) или ДНК. Необязательно, нуклеиновая кислота, кодирующая белок Cas, может быть кодон-оптимизирована для эффективной трансляции в белок в конкретной клетке или организме. Например, нуклеиновая кислота, кодирующая белок Cas, может быть модифицирована с целью замещения кодонов, которые часто используются в клетке бактерии, клетке дрожжей, клетке человека, клетке организма, не являющегося человеком, клетке млекопитающего, клетке грызуна, клетке мыши, клетке крысы или любой другой представляющей интерес клетке-хозяине по сравнению с полинуклеотидной последовательностью, встречающейся в природе. Например, нуклеиновая кислота, кодирующая белок Cas, может быть кодон-оптимизирована для экспрессии в клетке человека. Когда нуклеиновая кислота, кодирующая белок Cas, вводится в клетку, белок Cas может кратковременно, условно или конститутивно экспрессироваться в клетке.
[00161] Белки Cas, представленные в виде иРНК, могут быть модифицированы для улучшения стабильности и (или) иммуногенных свойств. Модификации могут быть внесены в один или большее число нуклеозидов внутри иРНК. Примеры химических модификаций нуклеиновых оснований иРНК включают в себя псевдоуридин, 1-метил-псевдоуридин и 5-метилцитидин. Например, можно использовать кэпированную и полиаденилированную иРНК Cas, содержащую N1-метилпсевдоуридин. Точно так же иРНК Cas могут быть модифицированы за счет истощения уридина с использованием синонимичных кодонов.
[00162] Нуклеиновые кислоты, кодирующие белки Cas, могут быть стабильно интегрированы в геном клетки и функционально связаны с активным в клетке промотором. В одном конкретном примере нуклеиновая кислота, кодирующая белок Cas, может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 22 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 22, необязательно при этом данная нуклеиновая кислота кодирует белок, содержащий, состоящий по существу из, или состоящий из последовательности с SEQ ID NO: 21. В другом конкретном примере нуклеиновая кислота, кодирующая химерный белок Cas, содержащий белок Cas с инактивированной нуклеазной активностью и один или большее число доменов активации транскрипции, может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 38 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 38, необязательно при этом данная нуклеиновая кислота кодирует белок, содержащий, состоящий по существу из, или состоящий из последовательности с SEQ ID NO: 36. В качестве альтернативы, нуклеиновые кислоты, кодирующие белки Cas, могут быть функционально связаны с промотором в экспрессионной конструкции. Экспрессионные конструкции включают в себя любые конструкции нуклеиновых кислот, способные регулировать экспрессию гена или другой представляющей интерес последовательности нуклеиновой кислоты (например, гена Cas) и которые могут переносить такую представляющую интерес последовательность нуклеиновой кислоты в клетку-мишень. Промоторы, которые можно использовать в экспрессионной конструкции, включают в себя промоторы, активные, например, в одной или большем числе из эукариотических клеток, клеток человека, негуманизированных клеток, клеток млекопитающего, негуманизированных клеток млекопитающего, клеток грызунов, клеток мыши, клеток крысы, плюрипотентных клеток, эмбриональных стволовых (ЭС) клеток, взрослых стволовых клеток, клеток-предшественниц с ограниченным развитием, индуцированных плюрипотентных стволовых (иПС, англ. «iPS») клеток или эмбриона на одноклеточной стадии. Такие промоторы могут представлять собой, например, условные промоторы, индуцируемые промоторы, конститутивные промоторы или тканеспецифические промоторы.
3. Химерные адаптерные белки
[00163] Описанные в данном документе биосенсорные клетки SAM/тау могут содержать не только нуклеиновые кислоты (ДНК или РНК), кодирующие химерный белок Cas, который содержит белок Cas с инактивированной нуклеазной активностью, слитый с одним или большим числом доменов активации транскрипции (например, VP64), но, необязательно, могут также содержать нуклеиновые кислоты (ДНК или РНК), кодирующие химерный адаптерный белок, который содержит адаптерный белок (например, белок оболочки MS2 (МСР)), слитый с одним или большим числом доменов активации транскрипции (например, слитый с р65 и HSF1). Необязательно, указанные химерный белок Cas и (или) химерный адаптерный белок стабильно экспрессируются. Необязательно, указанные клетки содержат геномно интегрированную последовательность, кодирующую химерный белок Cas и (или) геномно интегрированную последовательность, кодирующую химерный адаптерный белок.
[00164] Такие химерные адаптерные белки включают в себя: (а) адаптер (т.е. адаптерный домен или адаптерный белок), который специфически связывается с адаптер-связывающим элементом в гидовой РНК; и (b) один или большее число гетерологичных доменов активации транскрипции. Например, такие слитые белки могут содержать 1, 2, 3, 4, 5 или большее число доменов активации транскрипции (например, два или большее число гетерологичных доменов активации транскрипции, или три или большее число гетерологичных доменов активации транскрипции). В одном из примеров такие химерные адаптерные белки могут включать в себя: (а) адаптер (т.е. адаптерный домен или адаптерный белок), который специфически связывается с адаптер-связывающим элементом в гидовой РНК; и (b) два или большее число доменов активации транскрипции. Например, химерный адаптерный белок может включать в себя: (а) адаптерный белок оболочки MS2, который специфически связывается с одним или большим числом аптаперов MS2 в гидовой РНК (например, с двумя аптамерами MS2 в разных областях в гидовой РНК); и (b) один или большее число (например, два или большее число) доменов активации транскрипции. Например, указанные два домена активации транскрипции могут представлять собой домены активации транскрипции р65 и HSF1 или их функциональные фрагменты или варианты. Тем не менее, также представлены химерные адаптерные белки, в которых домены активации транскрипции содержат другие домены активации транскрипции или их функциональные фрагменты или варианты.
[00165] Указанные один или большее число доменов активации транскрипции могут быть непосредственно слиты с адаптером. В качестве альтернативы, указанные один или большее число доменов активации транскрипции могут быть связаны с адаптером посредством линкера или комбинации линкеров, или посредством одного или большего числа дополнительных доменов. Аналогичным образом, если присутствуют два или большее число доменов активации транскрипции, то они могут быть непосредственно слиты друг с другом или могут быть связаны друг с другом посредством линкера или комбинации линкеров, или посредством одного или большего числа дополнительных доменов. Линкеры, которые можно использовать в указанных слитых белках, могут включать в себя любую последовательность, которая не влияет на функцию указанных слитых белков. Иллюстративные линкеры представляют собой короткие (например, состоящие из 2-20 аминокислот) и обычно гибкие (например, содержащие аминокислоты с высокой степенью свободы, такие как глицин, аланин и серии) линкеры.
[00166] Указанные один или большее число доменов активации транскрипции и указанный адаптер могут располагаться в любом порядке внутри указанного химерного адаптерного белка. В качестве одного из вариантов, указанные один или большее число доменов активации транскрипции могут быть С-концевыми по отношению к указанному адаптеру, а указанный адаптер может быть N-концевым по отношению к указанным одному или большему числу доменов активации транскрипции. Например, указанные один или большее число доменов активации транскрипции могут находиться на С-конце указанного химерного адаптерного белка, а указанный адаптер может находиться на N-конце указанного химерного адаптерного белка. Тем не менее, указанные один или большее число доменов активации транскрипции могут быть С-концевыми по отношению к указанному адаптеру, не находясь на С-конце указанного химерного адаптерного белка (например, если сигнал ядерной локализации находится на С-конце указанного химерного адаптерного белка). Аналогично, указанный адаптер может быть N-концевым по отношению к указанным одному или большему числу доменов активации транскрипции, не находясь на N-конце указанного химерного адаптерного белка (например, если сигнал ядерной локализации находится на N-конце указанного химерного адаптерного белка). В качестве другого варианта, указанные один или большее число доменов активации транскрипции могут быть N-концевыми по отношению к указанному адаптеру, а указанный адаптер может быть С-концевым по отношению к указанным одному или большему числу доменов активации транскрипции. Например, указанные один или большее число доменов активации транскрипции могут находиться на N-конце указанного химерного адаптерного белка, а указанный адаптер может находиться на С-конце указанного химерного адаптерного белка. В качестве еще одного варианта, если указанный химерный адаптерный белок содержит два или большее число доменов активации транскрипции, то указанные два или большее число доменов активации транскрипции могут фланкировать указанный адаптер.
[00167] Химерные адаптерные белки также могут быть функционально связаны или слиты с дополнительными гетерологичными полипептидами. Указанный слитый или связанный гетерологичный полипептид может быть расположен на N-конце, С-конце или в любой области внутри указанного химерного адаптерного белка. Например, химерный адаптерный белок может дополнительно содержать сигнал ядерной локализации. Конкретный пример такого белка включает в себя белок оболочки MS2 (адаптер), связанный (либо напрямую, либо посредством NLS) с доменом активации транскрипции р65, С-концевым по отношению к белку оболочки MS2 (МСР), и с доменом активации транскрипции HSF1, С-концевым по отношению к домену активации транскрипции р65. Такой белок может содержать, в направлении от N-конца до С-конца: белок МСР; сигнал ядерной локализации; домен активации транскрипции р65; и домен активации транскрипции HSF1. Например, химерный адаптерный белок может содержать, состоять по существу из, или состоять из аминокислотной последовательности, которая по меньшей мере на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности химерного адаптерного белка MCP-p65-HSF1, указанной в SEQ ID NO: 37. Аналогичным образом, нуклеиновая кислота, кодирующая химерный адаптерный белок, может содержать, состоять по существу из, или состоять из последовательности, которая по меньшей мере на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности, кодирующей химерный адаптерный белок MCP-p65-HSF1, указанной в SEQ ID NO: 39.
[00168] Адаптеры (т.е. адаптерные домены или адаптерные белки) представляют собой домены, связывающие нуклеиновые кислоты (например, ДНК-связывающие домены и (или) РНК-связывающие домены), которые специфически распознают и связываются с определенными последовательностями (например, связываются с определенными последовательностями ДНК и (или) РНК, такими как аптамеры, зависимым от последовательности способом). Аптамеры включают в себя нуклеиновые кислоты, которые, благодаря своей способности принимать определенную трехмерную конформацию, могут связываться с целевой молекулой с высокой аффинностью и специфичностью. Такие адаптеры могут связываться, например, с определенной последовательностью РНК и вторичной структурой. Данные последовательности (т.е. адаптер-связывающие элементы) могут быть сконструированы в гидовую РНК. Например, аптамер MS2 может быть сконструирован в гидовую РНК для специфического связывания белка оболочки MS2 (МСР). Например, указанный адаптер может содержать, состоять по существу из, или состоять из аминокислотной последовательности, которая по меньшей мере на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности МСР, указанной в SEQ ID NO: 40. Аналогичным образом, нуклеиновая кислота, кодирующая указанный адаптер, может содержать, состоять по существу из, или состоять из последовательности, которая по меньшей мере на 85%, на 90%, на 91%, на 92%, на 93%, на 94%, на 95%, на 96%, на 97%, на 98%, на 99% или на 100% идентична последовательности, кодирующей МСР, указанной в SEQ ID NO: 41. Конкретные примеры адаптеров и целевых молекул включают в себя, например, комбинации РНК-связывающий белок - аптамер, которые существуют в пределах разнообразия белков оболочки бактериофага. См., например, US 2019-0284572 и WO 2019/183123, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей.
[00169] Описанные в данном документе химерные адаптерные белки содержат один или большее число доменов активации транскрипции. Такие домены активации транскрипции могут представлять собой встречающиеся в природе домены активации транскрипции, могут представлять собой функциональные фрагменты или функциональные варианты встречающихся в природе доменов активации транскрипции, или могут представлять собой сконструированные или синтетические домены активации транскрипции. Домены активации транскрипции, которые можно применять, включают в себя домены, описанные, например, в US 2019-0284572 и WO 2019/183123, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей.
4. Типы клеток
[00170] Описанные в данном документе биосенсорные клетки Cas/тау могут представлять собой клетки любого типа и могут быть in vitro, ex vivo или in vivo. Линия биосенсорных клеток Cas/тау или популяция клеток может быть моноклональной клеточной линией или популяцией клеток. Аналогичным образом, описанные в данном документе биосенсорные клетки SAM/тау могут представлять собой клетки любого типа и могут быть in vitro, ex vivo или in vivo. Линия биосенсорных клеток SAM/тау или популяция клеток может быть моноклональной клеточной линией или популяцией клеток. Клетка может происходить из любого источника. Например, клетка может быть эукариотической клеткой, животной клеткой, растительной клеткой или грибковой (например, дрожжевой) клеткой. Такие клетки могут представлять собой клетки рыб или клетки птиц, или такие клетки могут представлять собой клетки млекопитающих, такие как клетки человека, клетки млекопитающих, не относящихся к человеку, клетки грызунов, клетки мыши или клетки крысы. Млекопитающие включают в себя, например, людей, приматов, не относящихся к человеку, человекообразных обезьян, нечеловекообразных обезьян, кошек, собак, лошадей, быков, оленей, бизонов, овец, грызунов (например, мышей, крыс, хомяков, морских свинок), сельскохозяйственных животных (например, виды бычьих, такие как коровы и быки; виды овечьих, такие как овцы и козы; и виды свинообразных, такие как свиньи и кабаны). Птицы включают в себя, например, кур, индюков, страусов, гусей и уток. Также включены домашние животные и сельскохозяйственные животные. Термин «животное, не являющееся человеком» исключает людей. В конкретном примере биосенсорные клетки Cas/тау представляют собой клетки человека (например, клетки НЕК293Т). Аналогичным образом, в конкретном примере биосенсорные клетки SAM/тау представляют собой клетки человека (например, клетки НЕК293Т).
[00171] Клетка может представлять собой, например, тотипотентную клетку или плюрипотентную клетку (например, эмбриональную стволовую (ЭС) клетку, такую как ЭС-клетка грызунов, ЭС-клетка мыши или ЭС-клетка крысы). Тотипотентные клетки включают в себя недифференцированные клетки, которые могут давать начало любому типу клеток, а плюрипотентные клетки включают в себя недифференцированные клетки, которые обладают способностью развиваться в больше чем один тип дифференцированных клеток. Такие плюрипотентные и (или) тотипотентные клетки могут представлять собой, например, ЭС-клетки или ЭС-подобные клетки, такие как индуцированные плюрипотентные стволовые (иПС) клетки. ЭС-клетки включают в себя тотипотентные или плюрипотентные клетки эмбрионального происхождения, которые способны вносить вклад в клеточную популяцию любой ткани развивающегося эмбриона при введении в эмбрион. ЭС-клетки могут происходить из внутренней клеточной массы бластоцисты и способны дифференцироваться в клетки любого из трех зародышевых листков позвоночных (энтодермы, эктодермы и мезодермы).
[00172] Клетка также может представлять собой первичную соматическую клетку или клетку, которая не является первичной соматической клеткой. Соматические клетки могут включать в себя любую клетку, которая не является гаметой, зародышевой клеткой, гаметоцитом или недифференцированной стволовой клеткой. Клетка может также представлять собой первичную клетку. Первичные клетки включают в себя клетки или культуры клеток, которые были выделены непосредственно из организма, органа или ткани. Первичные клетки включают в себя клетки, которые не являются ни трансформированными, ни бессмертными. Они включают в себя любую клетку, полученную из организма, органа или ткани, которая ранее не пересаживалась как культура клеток или ранее пересаживалась как культура клеток, но не может пересаживаться как культура клеток неопределенно долго. Такие клетки могут быть выделены с помощью обычных методик и включают в себя, например, соматические клетки, гематопоэтические клетки, эндотелиальные клетки, эпителиальные клетки, фибробласты, мезенхимальные клетки, кератиноциты, меланоциты, моноциты, мононуклеарные клетки, адипоциты, преадипоциты, нейроны, глиальные клетки, гепатоциты, скелетные миобласты и гладкомышечные клетки. Например, первичные клетки могут происходить из соединительных тканей, мышечных тканей, тканей нервной системы или эпителиальных тканей.
[00173] Также, такие клетки, как правило, не пролиферируют неопределенно долго, но из-за мутации или изменения избежали нормального клеточного старения и вместо этого могут продолжают делиться. Такие мутации или изменения могут происходить естественным образом или быть вызваны намеренно. Примеры иммортализованных клеток включают в себя клетки яичника китайского хомячка (СНО), эмбриональные клетки почки человека (например, клетки НЕК293Т) и клетки-фибробасты эмбриона мыши (например, клетки 3Т3). Многочисленные типы иммор тали зов анных клеток хорошо известны. Иммортализованные или первичные клетки включают в себя клетки, которые обычно используются для культивирования или для экспрессии рекомбинантных генов или белков.
[00174] Клетка может также представлять собой дифференцированную клетку, такую как нейрональная клетка (например, нейрональная клетка человека).
В. Способы получения биосенсорных клеток Cas/тау и биосенсорных клеток SAM/тау
[00175] Описанные в данном документе биосенсорные клетки Cas/тау могут быть получены любыми известными способами. Первый содержащий повторы домен тау-белка, связанный с первым репортером, второй содержащий повторы домен тау-белка, связанный со вторым репортером, и белок Cas могут быть введены в клетку в любом виде (например, ДНК, РНК или белок) любыми известными способами. Аналогичным образом, описанные в данном документе биосенсорные клетки SAM/тау могут быть получены любыми известными способами. Первый содержащий повторы домен тау-белка, связанный с первым репортером, второй содержащий повторы домен тау-белка, связанный со вторым репортером, химерный белок Cas и химерный адаптерный белок могут быть введены в клетку в любом виде (например, ДНК, РНК или белок) любыми известными способами. «Введение» включает в себя презентацию клетке нуклеиновой кислоты или белка таким образом, что соответствующая последовательность получает доступ внутрь клетки. Представленные в данном документе способы не зависят от конкретного способа введения нуклеиновой кислоты или белка в клетку, только от того, что данная нуклеиновая кислота или данный белок получают доступ внутрь по меньшей мере одной клетки. Способы введения нуклеиновых кислот и белков в различные типы клеток известны и включают в себя, например, способы стабильной трансфекции, способы кратковременной трансфекции и опосредованные вирусами способы. Необязательно, можно использовать нацеливающие векторы.
[00176] Протоколы трансфекции, а также протоколы введения нуклеиновых кислот или белков в клетки могут быть различными. Неограничивающие способы трансфекции включают в себя химические способы трансфекции с использованием липосом; наночастищ фосфата кальция (Graham et al. (1973) Virology 52 (2): 456-67, Bacchetti et al. (1977) Proc. Natl. Acad. Sci. USA 74 (4): 1590 - 4; и Kriegler, M (1991). Transfer and Expression: A Laboratory Manual. New York: W. H. Freeman and Company, pp. 96-97); дендримеров; или катионных полимеров, такие как диэтиламиноэтилдекстран или полиэтиленимин. Нехимические способы включают в себя электропорацию, сонопорацию и оптическую трансфекцию. Трансфекция на основе частиц включает в себя применение генной пушки или трансфекцию с помощью магнита (Bertram (2006) Current Pharmaceutical Biotechnology 7, 277 - 28). Для трансфекции также можно применять вирусные способы.
[00177] Введение нуклеиновых кислот или белков в клетку также может быть опосредовано электропорацией, интрацитоплазматической инъекцией, вирусной инфекцией, аденовирусом, аденоассоциированным вирусом, лентивирусом, ретровирусом, трансфекцией, липид-опосредованной трансфекцией или нуклеофекцией. Нуклеофекция представляет собой усовершенствованную технологию электропорации, которая позволяет доставлять субстраты нуклеиновых кислот не только в цитоплазму, но также через ядерную мембрану и в ядро. В дополнение к этому, для применения нуклеофекции в описанных в данном документе способах обычно требуется гораздо меньше клеток, чем при обычной электропорации (например, всего около 2 миллионов по сравнению с 7 миллионами при обычной электропорации). В одном из примеров нуклеофекция выполняется с использованием системы LONZA ® NUCLEOFECTOR™.
[00178] Введение нуклеиновых кислот или белков в клетку также можно осуществить с помощью микроинъекции. Микроинъекция иРНК предпочтительно осуществляется в цитоплазму (например, для доставки иРНК непосредственно в клеточную систему трансляции), тогда как микроинъекция белка или ДНК, кодирующей белок, предпочтительно осуществляется в ядро. В качестве альтернативы, микроинъекция может осуществляться с помощью инъекции как в ядро, так и в цитоплазму: сначала игла может быть введена в ядро, и может быть произведена инъекция первого количества, а при извлечении иглы из клетки может быть введено второе количество в цитоплазму. Способы проведения микроинъекций хорошо известны. См., например, работу Nagy et al. (Nagy A, Gertsenstein M, Vintersten K, Behringer R., 2003, Manipulating the Mouse Embryo. Cold Spring Harbor, New York: Cold Spring Harbor Laboratory Press); работу Meyer et al. (2010) Proc. Natl. Acad. Sci. USA 107: 15022 - 15026; и работу Meyer et al. (2012) Proc. Natl. Acad. Sci. USA 109: 9354 - 9359.
[00179] Другие способы введения нуклеиновой кислоты или белков в клетку могут включать в себя, например, векторную доставку, опосредованную частицами доставку, опосредованную экзосомами доставку, опосредованную липидными наночастицами доставку, опосредованную проникающим в клетку пептидом доставку или опосредованную имплантируемым устройством доставку. Способы введения нуклеиновых кислот или белков субъекту для модификации клеток in vivo описаны в другой части данного документа.
[00180] В одном примере первый содержащий повторы домен тау-белка, связанный с первым репортером, второй содержащий повторы домен тау-белка, связанный со вторым репортером, и белок Cas могут быть введены с помощью вирусной трансдукции, такой как лентивирусная трансдукция.
[00181] Скрининг клеток, которые содержат первый содержащий повторы домен тау-белка, связанный с первым репортером, второй содержащий повторы домен тау-белка, связанный со вторым репортером, и белок Cas, можно проводить любыми известными способами.
[00182] В качестве одного из примеров, можно применять репортерные гены для скрининга клеток, которые содержат белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, или второй содержащий повторы домен тау-белка, связанный со вторым репортером. Иллюстративные репортерные гены включают в себя гены, кодирующие люциферазу, β-галактозидазу, зеленый флуоресцентный белок (GFP), усиленный зеленый флуоресцентный белок (eGFP), голубой флуоресцентный белок (CFP), желтый флуоресцентный белок (YFP), усиленный желтый флуоресцентный белок (eYFP), синий флуоресцентный белок (BFP), усиленный синий флуоресцентный белок (eBFP), DsRed, ZsGreen, MmGFP, mPlum, mCherry, tdTomato, mStrawberry, J-Red, mOrange, mKO, mCitrine, Venus, YPet, Emerald, CyPet, Cerulean, T-Sapphire и щелочную фосфатазу. Например, если первый репортер и второй репортер представляют собой флуоресцентные белки (например, CFP и YFP), то клетки, содержащие данные репортеры, могут быть отобраны с помощью технологии проточной цитометрии для отбора дважды положительных клеток. Затем данные дважды положительные клетки могут быть объединены для получения поликлональной линии, или могут быть получены моноклональные линии из одиночных дважды положительных клеток.
[00183] В качестве одного из примеров, можно применять маркеры отбора для скрининга клеток, которые содержат белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, или второй содержащий повторы домен тау-белка, связанный со вторым репортером. Примеры маркеров отбора включают в себя неомицинфосфотрансферазу (neor), гигромицин-В-фосфотрансферазу (hygr), пуромицин-N-ацетилтрансферазу (puror), бластицидин-S-дезаминазу (bsrr), ксантин-гуанинфосфорибозилтрансферазу (gpt) или тимидинкиназу вируса простого герпеса (HSV-k). Другой иллюстративный маркер отбора представляет собой белок устойчивости к блеомицину, кодируемый геном Sh ble (ген блеомицина из Streptoalloteichus hindustanus), который придает устойчивости к зеоцину (флеомицину D1).
[00184] Положительные в отношении агрегации тау-белка (Аг[+]) клетки, в которых содержащий повторы домен тау-белка стабильно присутствует в агрегированном состоянии, что означает, что агрегаты содержащего повторы домена тау-белка стабильно сохраняются во всех клетках при росте и множественных пассажах со временем, могут быть получены, например, путем засева агрегатами тау-белка. Например, наивные отрицательные в отношении агрегации тау-белка (Аг[-]) биосенсорные клетки Cas/тау, описанные в данном документе, могут быть обработаны рекомбинантным фибриллизованным тау-белком (например, рекомбинантным фибриллизованным доменом тау-белка, содержащим повторы) для засева агрегации содержащего повторы домена тау-белка, стабильно экспрессируемого данными клетками. Аналогичным образом, наивные отрицательные в отношении агрегации тау-белка (Аг[-]) биосенсорные клетки SAM/тау, описанные в данном документе, могут быть обработаны рекомбинантным фибриллизованным тау-белком (например, рекомбинантным фибриллизованным доменом тау-белка, содержащим повторы) для засева агрегации содержащего повторы домена тау-белка, стабильно экспрессируемого данными клетками. Фибриллизованный содержащий повторы домен тау-белка может быть таким же, как содержащий повторы домен тау-белка, стабильно экспрессируемый клетками, или отличаться от него. Необязательно, рекомбинантный фибриллизованный тау-белок может быть смешан с липофектаминовым реагентом. Затем засеянные клетки могут быть серийно разведены для получения клонов-производных от одиночных клеток и для идентификации клональных линий клеток, в которых агрегаты содержащих повторы доменов тау-белка стабильно сохраняются во всех клетках при росте и множественных пассажах с течением времени.
С. Культуры in vitro
[00185] В данном документе также описаны культуры in vitro или композиции, содержащие описанные в данном документе биосенсорные клетки Cas/тау и среду для культивирования указанных клеток. В данном документе также описаны культуры in vitro или композиции, содержащие описанные в данном документе биосенсорные клетки SAM/тау и среду для культивирования указанных клеток. Данные клетки могут представлять собой клетки Аг[-] или клетки Аг[+].
III. Библиотеки гидовых РНК для нокаута генов
[00186] В способах скрининга CRISPRn, описанных в данном документе, применяются библиотеки CRISPR гидовых РНК (гРНК) для нокаута генов, такие как полногеномные библиотеки гРНК для нокаута генов. Нуклеазы Cas, такие как Cas9, могут быть запрограммированы на индукцию двухцепочечных разрывов в определенных геномных локусах с помощью гРНК, предназначенных для нацеливания на определенные целевые последовательности. Поскольку специфичность нацеливания белков Cas обеспечивается короткими гРНК, возможен объединенный функциональный скрининг в масштабе генома. Такие библиотеки имеют несколько преимуществ перед другими библиотеками, такими как библиотеки коротких шпилечных РНК (кшРНК, англ. «shRNA»), которые снижают экспрессию белка за счет нацеливания на иРНК. Напротив, библиотеки гРНК достигают нокаута за счет мутаций сдвига рамки считывания, введенных в геномные кодирующие области генов.
[00187] В способах скрининга CRISPRa, описанных в данном документе, применяются библиотеки CRISPR гидовых РНК (гРНК) для активации транскрипции, такие как полногеномные библиотеки гРНК для активации транскрипции. Системы SAM могут быть запрограммированы на активацию транскрипции генов в определенных геномных локусах с помощью гРНК, предназначенных для нацеливания на определенные целевые последовательности. Поскольку специфичность нацеливания белков Cas обеспечивается короткими гРНК, возможен объединенный функциональный скрининг в масштабе генома.
[00188] гРНК в библиотеке могут быть нацелены на любое число генов. Например, гРНК могут быть нацелены на около 50 или большее число генов, на около 100 или большее число генов, на около 200 или большее число генов, на около 300 или большее число генов, на около 400 или большее число генов, на около 500 или большее число генов, на около 1000 или большее число генов, на около 2000 или большее число генов, на около 3000 или большее число генов, на около 4000 или большее число генов, на около 5000 или большее число генов, на около 10000 или большее число генов, или на около 20000 или большее число генов. В некоторых библиотеках гРНК могут быть выбраны для нацеливания на гены в конкретном сигнальном пути. Некоторые библиотеки представляют собой полногеномные библиотеки.
[00189] Полногеномные библиотеки включают в себя одну или большее число гРНК (например, егРНК), нацеленных на каждый ген в геноме. Целевой геном может представлять собой геном любого типа. Например, геном может представлять собой эукариотический геном, геном млекопитающего, геном млекопитающего, отличного от человека, геном грызунов, геном мыши, геном крысы или геном человека. В одном из примеров целевой геном представляет собой геном человека.
[00190] гРНК могут быть нацелены на любое число последовательностей в каждом отдельном целевом гене. В некоторых библиотеках нацеливание происходит в среднем на множество целевых последовательностей в каждом из целевого множества генов. Например, нацеливание происходит в среднем на от около 2 до около 10, от около 2 до около 9, от около 2 до около 8, от около 2 до около 7, от около 2 до около 6, от около 2 до около 5, от около 2 до около 4 или от около 2 до около 3 уникальных целевых последовательностей в каждом из целевого множества генов. Например, нацеливание происходит в среднем на по меньшей мере около 2, на по меньшей мере около 3, на по меньшей мере около 4, на по меньшей мере около 5 или на по меньшей мере около 6 уникальных целевых последовательностей в каждом из целевого множества генов. В качестве конкретного примера, нацеливание происходит в среднем на около 6 целевых последовательностей в каждом из целевого множества генов. В качестве другого конкретного примера, нацеливание происходит в среднем на от около 3 до около 6 или на от около 4 до около 6 целевых последовательностей в каждом из целевого множества генов.
[00191] Например, библиотеки могут быть нацелены на гены со средним охватом около 1, около 2, около 3, около 4, около 5, около 6, около 7, около 8, около 9 или около 10 гРНК на ген. В конкретном примере библиотека может быть нацелена на гены со средним охватом около 3-4 гРНК на ген или около 6 гРНК на ген.
[00192] гРНК могут быть нацелены на любую желаемую область в целевых генах. гРНК CRISPRn могут быть сконструированы для нацеливания на кодирующие области генов таким образом, что расщепление соответствующим белком Cas приведет к мутациям вставки/делеции (В/Д) со сдвигом рамки считывания, которые приводят к образованию аллеля с потерей функции. Более конкретно, мутации со сдвигом рамки считывания могут быть достигнуты с помощью нацеленных разрывов двухцепочечных ДНК и последующей мутагенной репарации через путь негомологичного соединения концов (НГСК), который приводит к образованию В/Д в месте разрыва. В/Д, вводимые в разрывы двухцепочечных ДНК, являются случайными мутациями, при этом некоторые В/Д приводят к мутациям со сдвигом рамки считывания, которые вызывают преждевременную терминацию соответствующего гена.
[00193] В некоторых библиотеках CRISPRn каждая гРНК нацелена на конститутивный экзон, если это возможно. В некоторых библиотеках CRISPRn каждая гРНК нацелена на 5'-конститутивный экзон, если это возможно. В некоторых способах каждая гРНК нацелена на первый экзон, второй экзон или третий экзон (с 5'-конца гена), если это возможно.
[00194] В качестве одного из примеров, гРНК в библиотеке CRISPRn могут быть нацелены на конститутивные экзоны. Конститутивные экзоны представляют собой экзоны, которые неизменно сохраняются после сплайсинга. Экзоны, экспрессируемые во всех тканях, можно рассматривать как конститутивные экзоны для нацеливания гРНК. гРНК в библиотеке могут быть нацелены на конститутивные экзоны вблизи 5'-конца каждого гена. Необязательно, первый и последний экзоны каждого гена могут быть исключены из потенциальных мишеней. Необязательно, любой экзон, содержащий сайт альтернативного сплайсинга, может быть исключен из потенциальных мишеней. Необязательно, два наиболее ранних экзона, отвечающих указанным выше критериям, выбираются в качестве потенциальных мишеней. Необязательно, экзоны 2 и 3 выбираются в качестве потенциальных мишеней (например, если конститутивные экзоны не идентифицированы). Кроме того, гРНК в библиотеке могут быть выбраны и сконструированы таким образом, чтобы минимизировать нецелевые эффекты.
[00195] В конкретном примере, полногеномная библиотека или библиотеки гРНК CRISPRn содержат егРНК, нацеленные на 5'-конститутивные экзоны >18000 генов в геноме человека со средним охватом ~ 6 егРНК на ген, при этом каждый целевой сайт был выбран с целью минимизации нецелевой модификации.
[00196] гРНК CRISPRa могут быть сконструированы для нацеливания на последовательности, примыкающие к сайту начала транскрипции гена. Например, целевая последовательность может быть расположена в пределах 1000, 900, 800, 700, 600, 500, 400, 300, 200, 190, 180, 170, 160, 150, 140, 130, 120, 110, 100, 90, 80, 70, 60, 50, 40, 30, 20, 10, 5 или 1 пары оснований от сайта начала транскрипции. Например, каждая гРНК в библиотеке CRISPRa может быть нацелена на последовательность в пределах 200 п. о. в направлении к 5' от сайта начала транскрипции. Необязательно, целевая последовательность находится в пределах области из 200 пар оснований в направлении к 5' от сайта начала транскрипции и 1 пары оснований направлении к 3' от сайта начала транскрипции (от -200 до +1).
[00197] гРНК в полногеномной библиотеке могут находиться в любом виде. Например, библиотека гРНК может быть упакована в вирусный вектор, такой как ретровирусные векторы, лентивирусные векторы или аденовирусные векторы. В конкретном примере библиотека гРНК упакована в лентивирусные векторы. Векторы могут дополнительно включать в себя репортерные гены или маркеры отбора для облегчения отбора клеток, которые получают векторы. Примеры таких репортерных генов и маркеров отбора описаны в другой части данного документа. В качестве одного из примеров, маркер отбора может представлять собой маркер, который придает устойчивость к лекарственному средству, такой как неомицинфосфотрансфераза, гигромицин-В-фосфотрансфераза, пуромицин-N-ацетилтрансфераза и бластицидин-S-дезаминаза. Другой иллюстративный маркер отбора представляет собой белок устойчивости к блеомицину, кодируемый геном Sh blе (ген блеомицина из Streptoalloteichus hindustanus), который придает устойчивости к зеоцину (флеомицину D1). Например, клетки можно отбирать с помощью лекарственного средства (например, пуромицина), таким образом, что только клетки, трансдуцированные конструкцией гидовой РНК, сохраняются для дальнейшего применения в проведении скрининга.
А. Гидовые РНК
[00198] «Гидовая РНК», или «гРНК», представляет собой молекулу РНК, которая связывается с белком Cas (например, с белком Cas9) и направляет белок Cas в определенную область внутри целевой ДНК. Гидовые РНК могут содержать два сегмента: «сегмент нацеливания на ДНК» и «сегмент связывания с белком». «Сегмент» включает в себя часть или область молекулы, такую как непрерывный участок нуклеотидов в РНК. Некоторые гРНК, например, гРНК для Cas9, могут содержать две отдельные молекулы РНК: «активирующую РНК» (например, тракрРНК) и «нацеливающую РНК» (например, CRISPR РНК или крРНК). Другие гРНК представляют собой одну молекулу РНК (полинуклеотид одной РНК), которую также можно назвать «одномолекулярной гРНК», «единой гидовой РНК» или «егРНК». См., например, патентные документы WO 2013/176772, WO 2014/065596, WO 2014/089290, WO 2014/093622, WO 2014/099750, WO 2013/142578 и WO 2014/131833, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. Для Cas9, например, единая гидовая РНК может содержать крРНК, слитую с тракрРНК (например, с помощью линкера). Для Cpfl, например, необходима только крРНК для достижения связывания с целевой последовательностью. Термины «гидовая РНК» и «гРНК» включают в себя как двухмолекулярные (т.е. модульные) гРНК, так и одномолекулярные гРНК.
[00199] Иллюстративная двухмолекулярная гРНК содержит крРНК-подобную («CRISPR РНК» или «нацеливающую РНК», или «крРНК», или «крРНК-повтор») молекулу и соответствующую тракрРНК-подобную («трансактивирующую CRISPR РНК» или «активирующую РНК», или «тракрРНК») молекулу. крРНК содержит как сегмент (одноцепочечный) гРНК для нацеливания на ДНК, так и участок нуклеотидов, который формирует половину дуплекса дцРНК сегмента гРНК для связывания с белком. Пример хвоста крРНК, расположенного в направлении к 3' от сегмента нацеливания на ДНК, включает в себя, состоит по существу из, или состоит из GUUUUAGAGCUAUGCU (SEQ ID NO: 23). Любой из описанных в данном документе сегментов нацеливания на ДНК может быть присоединен к 5'-концу SEQ ID NO: 23 с образованием крРНК.
[00200] Соответствующая тракрРНК (активирующая РНК) содержит отрезок нуклеотидов, который формирует другую половину дуплекса дцРНК сегмента гРНК для связывания с белком. Участок нуклеотидов крРНК комплементарен участку нуклеотидов тракрРНК и гибридизуется с участком нуклеотидов тракрРНК с образованием дуплекса дцРНК сегмента гРНК для связывания с белком. Таким образом, можно сказать, что каждая крРНК имеет соответствующую тракрРНК. Иллюстративная последовательность тракрРНК содержит, состоит по существу из, или состоит из
AGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGA GUCGGUGCUUU (SEQ ID NO: 24). Другие иллюстративные последовательности тракрРНК содержат, состоят по существу из, или состоят из любого из AAACAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUUUU (SEQ ID NO: 28) или
GUUGGAACCAUUCAAAACAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (SEQ ID NO: 29).
[00201] В системах, в которых необходимы как крРНК, так и тракрРНК, крРНК и соответствующая тракрРНК гибридизуются с образованием гРНК. В системах, в которых необходима только крРНК, крРНК может представлять собой гРНК. крРНК дополнительно обеспечивает одноцепочечный сегмент нацеливания на ДНК, который гибридизуется с комплементарной цепью целевой ДНК. При использовании для модификации внутри клетки, точная последовательность данной молекулы крРНК или тракрРНК может быть разработана так им образом, чтобы быть специфичной для видов, в которых будут использоваться данные молекулы РНК. См., например, работу Mali et al. (2013) Science 339: 823 - 826; работу Jinek et al. (2012) Science 337: 816 - 821; работу Hwang et al. (2013) Abrf. Biotechnol. 31: 227 - 229; работу Jiang et al. (2013)Nat. Biotechnol. 31: 233 - 239; и работу Cong et al. (2013) Science 339: 819 - 823, каждая из которых включена в данный документ посредством ссылки во всей своей полноте для всех целей.
[00202] Сегмент нацеливания на ДНК (крРНК) данной гРНК содержит нуклеотидную последовательность, которая комплементарна последовательности на комплементарной цепи целевой ДНК, как более подробно описано ниже. Сегмент гРНК для нацеливания на ДНК взаимодействует с целевой ДНК специфическим для последовательности образом посредством гибридизации (т.е. спаривания оснований). Как таковая, нуклеотидная последовательность сегмента нацеливания на ДНК может варьировать и определяет местоположение внутри целевой ДНК, в котором будут взаимодействовать гРНК и целевая ДНК. Сегмент данной гРНК для нацеливания на ДНК может быть модифицирован для гибридизации с любой желаемой последовательностью в целевой ДНК. Встречающиеся в природе крРНК различаются в зависимости от системы CRISPR/Cas и организма, но часто содержат сегмент нацеливания длиной от 21 нуклеотида до 72 нуклеотидов, фланкированный двумя прямыми повторами (ПП) длиной от 21 нуклеотида до 46 нуклеотидов (см., например, патентный документ WO 2014/131833, который включен в данный документ посредством ссылки во всей своей полноте для всех целей. В случае S. pyogenes, длина ПП составляет 36 нуклеотидов, а длина сегмента нацеливания составляет 30 нуклеотидов. Расположенный в 3'-направлении ПП комплементарен соответствующей тракрРНК и гибридизуется с соответствующей тракрРНК, которая в свою очередь связывается с белком Cas.
[00203] Сегмент нацеливания на ДНК может иметь, например, длину по меньшей мере около 12, 15, 17, 18, 19, 20, 25, 30, 35 или 40 нуклеотидов. Такие сегменты нацеливания на ДНК могут иметь, например, длину от около 12 нуклеотидов до около 100 нуклеотидов, от около 12 нуклеотидов до около 80 нуклеотидов, от около 12 нуклеотидов до около 50 нуклеотидов, от около 12 нуклеотидов до около 40 нуклеотидов, от около 12 нуклеотидов до около 30 нуклеотидов, от около 12 нуклеотидов до около 25 нуклеотидов или от около 12 нуклеотидов до около 20 нуклеотидов. Например, сегмент нацеливания на ДНК может состоять из от около 15 нуклеотидов до около 25 нуклеотидов (например, из от около 17 нуклеотидов до около 20 нуклеотидов, или из около 17 нуклеотидов, 18 нуклеотидов, 19 нуклеотидов или 20 нуклеотидов). См., например, патентный документ US 2016/0024523, который включен в данный документ посредством ссылки во всей своей полноте для всех целей. Для Cas9 из S. pyogenes типичный сегмент нацеливания на ДНК составляет от 16 нуклеотидов до 20 нуклеотидов в длину или от 17 нуклеотидов до 20 нуклеотидов в длину. Для Cas9 из S. aureus типичный сегмент нацеливания на ДНК составляет от 21 нуклеотида до 23 нуклеотидов в длину. Для Cpf1 типичный сегмент нацеливания на ДНК составляет по меньшей мере 16 нуклеотидов в длину или по меньшей мере 18 нуклеотидов в длину.
[00204] ТракрРНК могут иметь любой вид (например, полноразмерные тракрРНК или активные частичные тракрРНК) и различную длину. Они могут включать в себя первичные транскрипты или процессированные формы. Например, тракрРНК (как часть единой гидовой РНК или как отдельная молекула, представляющая собой часть двухмолекулярной гРНК) могут содержать, состоять по существу из, или состоять из всей или части последовательности тракрРНК дикого типа (например, из около или больше чем из около 20, 26, 32, 45, 48, 54, 63, 67, 85 или большего числа нуклеотидов из последовательности тракрРНК дикого типа). Примеры последовательностей тракрРНК дикого типа из S. pyogenes включают в себя 171-нуклеотидную, 89-нуклеотидную, 75-нуклеотидную и 65-нуклеотидную версии. См., например, Deltcheva etal. (2011) Nature 471: 602-607; WO 2014/093661, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. Примеры тракрРНК в единых гидовых РНК (егРНК) включают в себя сегменты тракрРНК, обнаруживаемые в +48, +54, +67 и +85 версиях егРНК, где «+n» указывает, что в егРНК включены нуклеотиды из тракрРНК дикого типа вплоть до +n нуклеотида из тракрРНК дикого типа. См. патентный документ US 8697359, который включен в данный документ посредством ссылки во всей своей полноте для всех целей.
[00205] Процент комплементарности между сегментом гидовой РНК для нацеливания на ДНК и комплементарной цепью целевой ДНК может составлять по меньшей мере 60% (например, по меньшей мере 65%, по меньшей мере 70%, по меньшей мере 75%, по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90%, по меньшей мере 95%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или по меньшей мере 100%). Процент комплементарности между сегментом нацеливания на ДНК и комплементарной цепью целевой ДНК может составлять по меньшей мере 60% в отношении около 20 смежных нуклеотидов. Например, процент комплементарности между сегментом нацеливания на ДНК и комплементарной цепью целевой ДНК может составлять 100% в отношении 14 смежных нуклеотидов на 5'-конце данной комплементарной цепи целевой ДНК и вплоть до 0% в отношении остальных нуклеотидов. В таком случае можно считать, что сегмент нацеливания на ДНК составляет 14 нуклеотидов в длину. В качестве другого примера, процент комплементарности между сегментом нацеливания на ДНК и комплементарной цепью целевой ДНК может составлять 100% в отношении семи смежных нуклеотидов на 5'-конце данной комплементарной цепи целевой ДНК и вплоть до 0% в отношении остальных нуклеотидов. В таком случае можно считать, что сегмент нацеливания на ДНК составляет 7 нуклеотидов в длину. В некоторых гидовых РНК по меньшей мере 17 нуклеотидов в сегменте нацеливания на ДНК комплементарны комплементарной цепи целевой ДНК. Например, сегмент нацеливания на ДНК может составлять 20 нуклеотидов в длину и может содержать 1, 2 или 3 несовпадения с комплементарной цепью целевой ДНК. В одном из примеров несовпадения не являются смежными с областью комплементарной цепи, соответствующей последовательности мотива, примыкающего к протоспейсеру (РАМ) (т.е. с обратным комплементом последовательности РАМ) (например, несовпадения находятся на 5'-конце сегмента гидовой РНК для нацеливания на ДНК, или несоответствия находятся на расстоянии по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18 или 19 пар оснований от области на комплементарной цепи, соответствующей последовательности РАМ).
[00206] Белок-связывающий сегмент гРНК может содержать два участка нуклеотидов, которые комплементарны друг другу. Комплементарные нуклеотиды белок-связывающего сегмента гибридизуются с образованием дуплекса двухцепочечной РНК (дцРНК). Сегмент данной гРНК для связывания с белком взаимодействует с белком Cas, и гРНК направляет связанный белок Cas к определенной нуклеотидной последовательности в целевой ДНК с помощью сегмента нацеливания на ДНК.
[00207] Единые гидовые РНК могут содержать сегмент нацеливания на ДНК и последовательность остова (т.е. белок-связывающую последовательность гидовой РНК или Cas-связывающую последовательность гидовой РНК). Например, такие гидовые РНК могут иметь 5'-сегмент нацеливания на ДНК, соединенный с 3'-последовательностью остова. Иллюстративные последовательности остова содержат, состоят по существу или состоят из: GUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCU (версия 1; SEQ ID NO: 17);
GUUGGAACCAUUCAAAACAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (версия 2; SEQ ID NO: 18);
GUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (версия 3; SEQ ID NO: 19); и
GUUUAAGAGCUAUGCUGGAAACAGCAUAGCAAGUUUAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (версия 4; SEQ ID NO: 20). Другие иллюстративные последовательности остова содержат, состоят по существу или состоят из:
GUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUUUUUUU (версия 5; SEQ ID NO: 30);
GUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUUUU (версия 6; SEQ ID NO: 31); или
GUUUAAGAGCUAUGCUGGAAACAGCAUAGCAAGUUUAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUUUUUU (версия 7; SEQ ID NO: 32). Гидовые РНК, нацеленные на любую из описанных в данном документе целевых последовательностей для гидовых РНК, могут включать в себя, например, сегмент на 5'-конце гидовой РНК для нацеливания на ДНК, слитый с любой из иллюстративных последовательностей каркаса остова гидовой РНК на 3'-конце гидовой РНК. Это значит, что любой из описанных в данном документе сегментов нацеливания на ДНК может быть присоединен к 5'-концу любой из указанных выше последовательностей остова с образованием единой гидовой РНК (химерной гидовой РНК).
[00208] Гидовые РНК могут включать в себя модификации или последовательности, которые обеспечивают дополнительные желательные свойства (например, модифицированную или регулируемую стабильность; субклеточное нацеливание; отслеживание с помощью флуоресцентной метки; сайт связывания для белка или белкового комплекса и т.п.). Примеры таких модификаций включают в себя, например, 5'-кэп (например, кэп 7-метилгуанилата (m7G)); 3'-полиаденилированный хвост (т.е. 3'-поли(А) хвост); последовательность рибопереключателя (например, для обеспечения возможности регулируемой стабильности и (или) регулируемой доступности для белков и (или) белковых комплексов); последовательность контроля стабильности; последовательность, которая образует дуплекс дцРНК (т.е. шпильку); модификацию или последовательность, которые нацеливают РНК на субклеточную локализацию (например, ядро, митохондрии, хлоропласты и т.п.); модификацию или последовательность, которые обеспечивают отслеживание (например, прямая конъюгация с флуоресцентной молекулой, конъюгация с фрагментом, который облегчает флуоресцентное обнаружение, последовательность, которая обеспечивает возможность флуоресцентного обнаружения, и т.д.); модификацию или последовательность, которые обеспечивают сайт связывания для белков (например, белков, которые действуют на ДНК, включая активаторы транскрипции, репрессоры транскрипции, ДНК-метилтрансферазы, ДНК-деметилазы, гистонацетилтрансферазы, гистондеацетилазы и т.п.); и их комбинации. Другие примеры модификаций включают в себя сконструированные дуплексные шпилечные структуры, сконструированные выпуклые области, сконструированные 3'-шпильки дуплексной шпилечной структуры или любую их комбинацию. См., например, патентный документ US 2015/0376586, который включен в данный документ посредством ссылки во всей своей полноте для всех целей. Выпуклая область может представлять собой неспаренную областью нуклеотидов внутри дуплекса, состоящую из крРНК-подобной области и минимальной тракрРНК-подобной области. Выпуклая область может содержать на одной стороне дуплекса неспаренную область 5' - XXXY - 3', где X представляет собой любой пурин, a Y может представлять собой нуклеотид, который может образовывать неоднозначную пару с нуклеотидом из противоположной цепи, и на другой стороне дуплекса - неспаренную нуклеотидную область.
[00209] В некоторых случаях может применяться система активации транскрипции, содержащая слитый белок dCas9-VP64, спаренный с MS2-p65-HSF1. Гидовые РНК в таких системах могут быть сконструированы с последовательностями аптамера, присоединенными к тетрапетле егРНК и шпильке 2, предназначенным для связывания димеризованных белков оболочки бактериофага MS2. См., например, работу Konermann et al. (2015) Nature 517 (7536): 583-588, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей.
[00210] Немодифицированные нуклеиновые кислоты могут быть подвержены деградации. Экзогенные нуклеиновые кислоты могут также вызывать врожденный иммунный ответ. Модификации могут способствовать повышению стабильности и снижению иммуногенности. Гидовые РНК могут содержать модифицированные нуклеозиды и модифицированные нуклеотиды, включая, например, одно или большее число из следующего: (1) изменение или замена одного или обоих несвязывающих атомов кислорода из фосфата и (или) одного или большего числа связывающих атомов кислорода из фосфата в связи фосфодиэфирного остова; (2) изменение или замена компонента рибозного сахара, такие как изменение или замена 2'-гидроксила в рибозном сахаре; (3) замена фосфатного фрагмента дефосфорилированными линкерами; (4) модификация или замена встречающегося в природе азотистого основания; (5) замена или модификация рибозо-фосфатного остова; (6) модификация 3'-конца или 5'-конца олигонуклеотида (например, удаление, модификация или замена концевой фосфатной группы или конъюгация фрагмента); и (7) модификация сахара. Другие возможные модификации гидовой РНК включают в себя модификации или замену урацилов или полиурациловых трактов. См., например, WO 2015/048577 и US 2016/0237455, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. Похожие модификации могут быть внесены в нуклеиновые кислоты, кодирующие Cas, такие как иРНК Cas. Например, иРНК Cas могут быть модифицированы путем истощения уридина с использованием синонимичных ко донов.
[00211] В качестве одного из примеров, нуклеотиды на 5'- или 3'-конце гидовой РНК могут включать в себя фосфоротиоатные связи (например, данные основания могут иметь модифицированную фосфатную группу, которая является фосфоротиоатной группой). Например, гидовая РНК может включать в себя фосфоротиоатные связи между 2, 3 или 4 концевыми нуклеотидами на 5'- или 3'-конце гидовой РНК. В качестве другого примера, нуклеотиды на 5'- и (или) 3'-конце гидовой РНК могут иметь 2'-O-метильные модификации. Например, гидовая РНК может включать в себя 2-О-метильные модификации на 2, 3 или 4 концевых нуклеотидах на 5'- и (или) 3'-конце гидовой РНК (например, на 5'-конце). См., например, WO 2017/173054 А1 и Finn et al. (2018) Cell Rep.22 (9): 2227 - 2235, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. Другие возможные модификации более подробно описаны в других частях данного документа. В конкретном примере гидовая РНК включает в себя 2-О-метильные аналоги и 3'-фосфоротиоатные межнуклеотидные связи в первых трех 5'- и 3'-концевых остатках РНК. Такие химические модификации могут, например, придавать гидовым РНК повышенную стабильность и защиту от экзонуклеаз, позволяя им сохраняться в клетках дольше, чем немодифицированные гидовые РНК. Такие химические модификации также могут, например, защищать от врожденных внутриклеточных иммунных ответов, которые могут активно разрушать РНК или запускать иммунные каскады, ведущие к гибели клеток.
[00212] В некоторых гидовых РНК (например, единых гидовых РНК) по меньшей мере одна петля (например, две петли) гидовой РНК модифицированы с помощью вставки определенной последовательности РНК, которая связывается с одним или большим числом адаптеров (например, адаптерными белками или доменами). Такие адаптерные белки могут быть использованы для дальнейшего рекрутирования одного или большего числа гетерологичных функциональных доменов, таких как домены активации транскрипции (например, для применения в скрининге CRISPRa в биосенсорных клетках SAM/тау). Примеры слитых белков, содержащих такие адаптерные белки (т.е. химерные адаптерные белки), описаны в другой части данного документа. Например, петля связывания MS2 ggccAACAUGAGGAUCACCCAUGUCUGCAGggcc (SEQ ID NO: 33) может заменять нуклеотиды с +13 на +16 и нуклеотиды с +53 на +56 остова егРНК (каркаса), указанного в последовательности с SEQ ID NO: 17 или с SEQ ID NO: 19 (или в последовательности с SEQ ID NO: 30 или 31), или остова егРНК для системы CRISPR/Cas9 из S. pyogenes, описанной в WO 2016/049258 и Konermann et al. (2015)Nature 517 (7536): 583 - 588, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. См. также патентные документы US 2019-0284572 и WO 2019/183123, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей. Применяемая в данном документе нумерация гидовых РНК относится к нумерации нуклеотидов в последовательности остова гидовой РНК (т.е. последовательности, расположенной в направлении к 3' от сегмента гидовой РНК, нацеливающего на ДНК). Например, первый нуклеотид остова гидовой РНК пронумерован как +1, второй нуклеотид остова пронумерован как +2 и т.д. Остатки, соответствующие нуклеотидам от +13 до +16 в последовательности с SEQ ID NO: 17 или с SEQ ID NO: 19 (либо в последовательности с SEQ ID NO: 30 или 31), представляют собой последовательность петли в области, охватывающей нуклеотиды от +9 до +21 в последовательности с SEQ ID NO: 17 или с SEQ ID NO: 19 (либо в последовательности с SEQ ID NO: 30 или 31), область, называемую в данном документе тетрапетлей. Остатки, соответствующие нуклеотидам от +53 до +56 в последовательности с SEQ ID NO: 17 или с SEQ ID NO: 19 (либо в последовательности с SEQ ID NO: 30 или 31), представляют собой последовательность петли в области, охватывающей нуклеотиды от +48 до +61 в последовательности с SEQ ID NO: 17 или с SEQ ID NO: 19 (либо в последовательности с SEQ ID NO: 30 или 31), область, называемую в данном документе шпилькой 2. Другие шпилечные последовательности в последовательности с SEQ ID NO: 17 или с SEQ ID NO: 19 (либо в последовательности с SEQ ID NO: 30 или 31) включают в себя шпильку 1 (нуклеотиды от +33 до +41) и шпильку 3 (нуклеотиды от +63 до +75). Получаемая в результате структура представляет собой остов егРНК, в котором каждая из последовательностей тетрапетли и шпильки 2 заменена петлей связывания MS2. Тетрапетля и шпилька 2 выступают из белка Cas9 таким образом, что добавление петли связывания MS2 не должно влиять на какие-либо остатки Cas9. Кроме того, близость сайтов тетрапетли и шпильки 2 к ДНК, указывает на то, что локализация в данных местоположениях может приводить к высокой степени взаимодействия между ДНК и любым рекрутируемым белком, таким как активатор транскрипции. Таким образом, в некоторых егРНК нуклеотиды, соответствующие положениям с +13 по +16, и (или) нуклеотиды, соответствующие положениям с +53 по +56 остова гидовой РНК, указанные в SEQ ID NO: 17 или в SEQ ID NO: 19 (или в SEQ ID NO: 30 или 31), или соответствующие остатки при оптимальном выравнивании с любым из данных остовов/каркасов заменяются отдельными последовательностями РНК, способными связываться с одним или большим числом адаптерных белков или доменов. В качестве альтернативы или дополнения, адаптер-связывающие последовательности могут быть добавлены к 5'-концу или 3'-концу гидовой РНК. Иллюстративный остов гидовой РНК, содержащий петли связывания MS2 в областях тетрапетли и шпильки 2, может содержать, состоять по существу из, или состоять из последовательности, указанной в SEQ ID NO: 34. Иллюстративная общая единая гидовая РНК, содержащая петли связывания MS2 в областях тетрапетли и шпильки 2, может содержать, состоять по существу из, или состоять из последовательности, указанной в SEQ ID NO: 35.
[00213] Гидовые РНК могут быть предоставлены в любом виде. Например, гРНК может быть предоставлена в виде РНК, либо в виде двух молекул (отдельные крРНК и тракрРНК), либо в виде одной молекулы (егРНК), и, необязательно, в виде комплекса с белком Cas. гРНК также может быть предоставлена в виде ДНК, кодирующей гРНК. ДНК, кодирующая гРНК, может кодировать одну молекулу РНК (егРНК) или отдельные молекулы РНК (например, отдельные крРНК и тракрРНК). В последнем случае ДНК, кодирующая гРНК, может быть предоставлена как одна молекула ДНК или как отдельные молекулы ДНК, кодирующие крРНК и тракрРНК, соответственно.
[00214] Когда гРНК предоставляется в форме ДНК, гРНК может кратковременно, условно или конститутивно экспрессироваться в клетке. ДНК, кодирующие гРНК, могут быть стабильно интегрированы в геном клетки и функционально связаны с активным в клетке промотором. В качестве альтернативы, ДНК, кодирующие гРНК, могут быть функционально связаны с промотором в экспрессионной конструкции. Например, ДНК, кодирующая гРНК, может находиться в векторе, содержащем гетерологичную нуклеиновую кислоту, такую как нуклеиновая кислота, кодирующая белок Cas. В качестве альтернативы, она может находиться в векторе или плазмиде, отдельной от вектора, которые содержат нуклеиновую кислоту, кодирующую белок Cas. Промоторы, которые можно использовать в таких экспрессионных конструкциях, включают в себя промоторы, активные, например, в одной или большем числе из эукариотических клеток, клеток человека, негуманизированных клеток, клеток млекопитающего, негуманизированных клеток млекопитающего, клеток грызунов, клеток мыши, клеток крысы, плюрипотентных клеток, эмбриональных стволовых (ЭС) клеток, взрослых стволовых клеток, клеток-предшественниц с ограниченным развитием, индуцированных плюрипотентных стволовых (иПС) клеток или эмбриона на одноклеточной стадии. Такие промоторы могут представлять собой, например, условные промоторы, индуцируемые промоторы, конститутивные промоторы или тканеспецифические промоторы. Такие промоторы также могут представлять собой, например, двунаправленные промоторы. Конкретные примеры подходящих промоторов включают в себя: промотор РНК-полимеразы III, такой как промотор U6 человека, промотор U6 полимеразы III крысы или промотор U6 полимеразы III мыши.
[00215] В качестве альтернативы, гРНК могут быть получены различными другими способами. Например, гРНК могут быть получены путем транскрипции in vitro с использованием, например, РНК-полимеразы Т7 (см., например, патентные документы WO 2014/089290 и WO 2014/065596, каждый из которых включен в данный документ посредством ссылки во всей своей полноте для всех целей). Гидовые РНК также могут представлять собой синтетически произведенные молекулы, полученные с помощью химического синтеза. Например, гидовая РНК может быть химически синтезирована, чтобы включать в себя 2'-O-метильные аналоги и 3'-фосфоротиоатные межнуклеотидные связи в первых трех 5'- и 3'-концевых остатках РНК.
[00216] Гидовые РНК (или нуклеиновые кислоты, кодирующие гидовые РНК) могут находиться в композициях, содержащих одну или большее число гидовых РНК (например, 1, 2, 3, 4 или большее число гидовых РНК) и носитель, повышающий стабильность гидовой РНК (например, носитель, пролонгирующий период при заданных условиях хранения (например, -20°С, 4°С или температура окружающей среды), в течение которого продукты разложения остаются ниже порогового значения, например, ниже 0,5% от веса исходной нуклеиновой кислоты или белка; или повышающий стабильность in vivo). Неограничивающие примеры таких носителей включают в себя микросферы полилактидной кислоты (PLA), микросферы поли-D,L-лактидной-когликолевой кислоты) (PLGA), липосомы, мицеллы, обратные мицеллы, липидные кохлеаты и липидные микротрубочки. Такие композиции могут дополнительно содержать белок Cas, такой как белок Cas9, или нуклеиновую кислоту, кодирующую белок Cas.
В. Целевые последовательности для гидовых РНК
[00217] Целевые ДНК для гидовых РНК включают в себя последовательности нуклеиновых кислот, присутствующие в ДНК, с которыми будет связываться направленный на ДНК сегмент гРНК, при условии, что существуют достаточные условия для связывания. Подходящие условия для связывания ДНК/РНК включают в себя физиологические условия, обычно присутствующие в клетке. Другие подходящие условия для связывания ДНК/РНК (например, условия в бесклеточной системе) известны в данной области техники (см., например, работу Molecular Cloning: A Laboratory Manual, 3rd Ed. (Sambrook et al., Harbor Laboratory Press 2001), которая включена в данный документ посредством ссылки во всей своей полноте для всех целей). Цепь целевой ДНК, которая комплементарна гРНК и гибридизуется с гРНК, может быть названа «комплементарной цепью», а цепь целевой ДНК, которая комплементарна «комплементарной цепи» (и, следовательно, не комплементарна белку Cas или гРНК) может быть названа «некомплементарной цепью» или «матричной цепью».
[00218] Целевая ДНК включает в себя как последовательность на комплементарной цепи, с которой гибридизуется гидовая РНК, так и соответствующую последовательность на некомплементарной цепи (например, смежную с мотивом, примыкающим к протоспейсеру (РАМ)). Термин «целевая последовательность для гидовой РНК» при использовании в контексте данного документа относится конкретно к последовательности на некомплементарной цепи, соответствующей (т.е. являющейся обратным комплементом) последовательности, с которой гибридизуется гидовая РНК на комплементарной цепи. Это значит, что целевая последовательность для гидовой РНК относится к последовательности на некомплементарной цепи, смежной с РАМ (например, в направлении к 5' от РАМ в случае Cas9). Целевая последовательность для гидовой РНК эквивалентна нацеливающему на ДНК сегменту гидовой РНК, но с тиминами вместо урацилов. В качестве одного из примеров, целевая последовательность для гидовой РНК применительно к ферменту SpCas9 может относиться к последовательности, расположенной в направлении к 5' от 5'-NGG-3' РАМ на некомплементарной цепи. Гидовая РНК разработана таким образом, чтобы иметь комплементарность к комплементарной цепи целевой ДНК, где гибридизация между нацеливающим на ДНК сегментом гидовой РНК и комплементарной цепью целевой ДНК способствует образованию комплекса CRISPR. Полная комплементарность не обязательно требуется при условии, что комплементарность достаточна, чтобы вызвать гибридизацию и способствовать образованию комплекса CRISPR. Если гидовая РНК упоминается в данном документе как нацеленная на целевую последовательность для гидовой РНК, это означает, что данная гидовая РНК гибридизуется с последовательностью комплементарной цепи целевой ДНК, которая является обратным комплементом целевой последовательности для гидовой РНК на некомплементарной цепи.
[00219] Целевая последовательность ДНК или целевая последовательность для гидовой РНК может содержать любой полинуклеотид и может располагаться, например, в ядре или в цитоплазме клетки, или в органелле клетки, такой как митохондрия или хлоропласт.Целевая последовательность ДНК или целевая последовательность для гидовой РНК может представлять собой любую последовательность нуклеиновой кислоты, эндогенную или экзогенную для клетки. Целевая последовательность для гидовой РНК может представлять собой последовательность, кодирующую продукт гена (например, белок), или некодирующую последовательность (например, регуляторную последовательность), или может включать в себя и то, и другое.
[00220] Для систем CRISPRa и SAM может быть предпочтительным, чтобы целевая последовательность была смежной с сайтом начала транскрипции гена. Например, целевая последовательность может быть расположена в пределах 1000, 900, 800, 700, 600, 500, 400, 300, 200, 190, 180, 170, 160, 150, 140, 130, 120, 110, 100, 90, 80, 70, 60, 50, 40, 30, 20, 10, 5 или 1 пары оснований от сайта начала транскрипции. Необязательно, целевая последовательность находится в пределах области из 200 пар оснований в направлении к 5' от сайта начала транскрипции и 1 пары оснований направлении к 3' от сайта начала транскрипции (от -200 до+1).
[00221] Сайт-специфическое связывание и расщепление целевой ДНК белком Cas может происходить в местоположениях, определяемых как (i) комплементарноетью спаривания оснований между гидовой РНК и комплементарной цепью целевой ДНК, так и (ii) коротким мотивом, называемым мотивом, примыкающим к протоспейсеру (РАМ), в некомплементарной цепи целевой ДНК. РАМ может фланкировать целевую последовательность для гидовой РНК. Необязательно, целевая последовательность для гидовой РНК может быть фланкирована последовательностью РАМ на 3'-конце (например, для Cas9). В качестве альтернативы, целевая последовательность для гидовой РНК может быть фланкирована последовательностью РАМ на 5'-конце (например, для Cpfl). Например, сайт расщепления для белков Cas может находиться на расстоянии от около 1 пары оснований до около 10 пар оснований или от около 2 пар оснований до около 5 пар оснований (например, 3 пары оснований) в направлении к 5' или в направлении к 3' от последовательности РАМ (например, в пределах целевой последовательности для гидовой РНК). В случае SpCas9, последовательность РАМ (т.е. на некомплементарной цепи) может представлять собой 5' - N1GG - 3', где Ni представляет собой любой нуклеотид ДНК, и где РАМ находится непосредственно в направлении к 3' от целевой последовательности для гидовой РНК на некомплементарной цепи целевой ДНК. Таким образом, последовательность, соответствующий РАМ на комплементарной цепи (т.е. обратный комплемент) будет представлять собой 5' - CCN2- 3', где N2 представляет собой любой нуклеотид ДНК и находится непосредственно в направлении к 5' от последовательности, с которой гибридизуется нацеливающий на ДНК сегмент гидовой РНК, на некомплементарной цепи целевой ДНК. В некоторых таких случаях N1 и N2 могут быть комплементарными, и пара оснований N1 - N2 может представлять собой любую пару оснований (например, N1=C и N2=G; N1=G и N2=C; N1=A и N2=T; или N1=T и N2=A). В случае Cas9 из S. aureus, последовательность РАМ может представлять собой NNGRRT или NNGRR, где N может представлять собой A, G, С или Т, a R может представлять собой G или А. В случае Cas9 из С.jejuni, последовательность РАМ может представлять собой, например, NNNNACAC или NNNNRYAC, где N может представлять собой A, G, С или Т, a R может представлять собой G или А. В некоторых случаях (например, для FnCpfl) последовательность РАМ может располагаться в направлении к 5' от 5'-конца и иметь последовательность 5' - TTN - 3'.
[00222] Примером целевой последовательности для гидовой РНК является последовательность ДНК из 20 нуклеотидов, непосредственно предшествующая мотиву NGG, которая распознается белком SpCas9. Например, двумя примерами целевых последовательностей для гидовой РНК плюс РАМ являются GN19NGG (последовательность с SEQ ID NO: 25) или N20NGG (последовательность с SEQ ID NO: 26). См., например, патентный документ WO 2014/165825, который включен в данный документ посредством ссылки во всей своей полноте для всех целей. Гуанин на 5'-конце может способствовать транскрипции РНК-полимеразой в клетках. Другие примеры целевых последовательностей для гидовой РНК плюс РАМ могут включать в себя два гуаниновых нуклеотида на 5'-конце (например, GGN20NGG; последовательность с SEQ ID NO: 27) для облегчения эффективной транскрипции полимеразой Т7 in vitro. См., например, патентный документ WO 2014/065596, который включен в данный документ посредством ссылки во всей своей полноте для всех целей. Другие целевые последовательности для гидовой РНК плюс РАМ могут иметь длину от 4 нуклеотидов до 22 нуклеотидов последовательностей с SEQ ID NO: 25-27, включительно с 5' G или GG и 3' GG или NGG. Еще другие целевые последовательности для гидовой РНК плюс РАМ могут иметь длину от 14 нуклеотидов до 20 нуклеотидов последовательностей с SEQ ID NO: 25-27.
[00223] Образование комплекса CRISPR, гибридизованного с целевой ДНК, может приводить к расщеплению одной или обеих цепей целевой ДНК внутри или вблизи области, соответствующей целевой последовательности для гидовой РНК (т.е. целевой последовательности для гидовой РНК на некомплементарной цепи целевой ДНК и обратному комплементу на комплементарной цепи, с которой гибридизуется гидовая РНК). Например, сайт расщепления может располагаться в последовательности-мишени направляющей РНК (например, в определенном месте относительно последовательности РАМ). «Сайт расщепления» включает в себя положение в целевой ДНК, в котором белок Cas вызывает одноцепочечный разрыв или двухцепочечный разрыв. Сайт расщепления может находиться только на одной цепи (например, когда используется никаза) или на обеих цепях двухцепочечной ДНК. Сайты расщепления могут находиться в одном и том же положении на обеих цепях (образуя тупые концы; например, Cas9)) или могут находиться в разных сайтах на каждой цепи (создавая ступенчатые концы (например, выступы); например, Cpfl). Ступенчатые концы могут быть получены, например, с помощью использования двух белков Cas, каждый из которых вызывает одноцепочечный разрыв в сайте расщепления, различном для одной и для другой цепи, тем самым вызывая двухцепочечный разрыв. Например, первая никаза может создавать одноцепочечный разрыв в первой цепи двухцепочечной ДНК (дцДНК), а вторая никаза может создавать одноцепочечный разрыв во второй цепи дцДНК, и, таким образом, создаются последовательности с выступами. В некоторых случаях целевая последовательность для гидовой РНК или сайт расщепления для никазы в первой цепи отделены от целевой последовательности для гидовой РНК или от сайта расщепления для никазы во второй цепи по меньшей мере на 2, на 3, на 4, на 5, на 6, на 7, на 8, на 9, на 10, на 15, на 20, на 25, на 30, на 40, на 50, на 75, на 100, на 250, на 500 или на 1000 пар оснований.
IV. Способы исключающего скрининга для выявления генетических уязвимостей, связанных с агрегацией тау-белка
[00224] Описанные в данном документе линии биосенсорных клеток Cas/тау могут применяться в способах исключающего скрининга для выявления генетических уязвимостей, связанных с агрегацией тау-белка. Такие способы могут включать в себя обеспечение положительных в отношении агрегации тау-белка и отрицательных в отношении агрегации тау-белка популяций биосенсорных клеток Cas/тау, как описано в другом месте в данном документе, введение библиотеки, содержащей множество уникальных гидовых РНК, в каждую в популяцию, и определение содержания (например, числа считываний) каждой из указанного множества уникальных гидовых РНК во множестве моментов времени в исследовании с течением времени в каждой популяции клеток.
[00225] В качестве одного из примеров, способ может включать в себя обеспечение положительной в отношении агрегации тау-белка популяции клеток и отрицательной в отношении агрегации тау-белка популяции клеток, при этом каждая популяция клеток содержит белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, как описано в другом месте в данном документе. В положительной в отношении агрегации тау-белка популяции клеток первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, присутствуют стабильно в агрегированном состоянии, тогда как в отрицательной в отношении агрегации тау-белка популяции клеток первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, не присутствуют стабильно в агрегированном состоянии. Способ может далее включать в себя введение в каждую популяцию клеток библиотеки, содержащей множество уникальных гидовых РНК, которые нацелены на множество генов. Множество уникальных гидовых РНК образует комплексы с белком Cas, и белок Cas расщепляет множество генов, что приводит к нокауту генной функции. Наконец, содержание каждой из множества уникальных гидовых РНК может быть определено во множестве моментов времени в исследовании с течением времени в каждой популяции клеток. Истощение гидовой РНК в положительной в отношении агрегации тау-белка популяции клеток, но не в отрицательной в отношении агрегации тау-белка популяции клеток, может указывать на то, что ген, на который нацелена данная гидовая РНК, проявляет синтетическую летальность с агрегатами тау-белка и является генетической уязвимостью, связанной с агрегацией тау-белка, или является потенциальной генетической уязвимостью, связанной с агрегацией тау-белка (например, кандидатом для дальнейшего тестирования с помощью вторичных скринингов), при этом ожидается, что нарушение гена, на который нацелена данная гидовая РНК, проявляет синтетическую летальность с агрегатами тау-белка. В качестве альтернативы, паттерн более резкого истощения гидовой РНК с течением времени в положительной в отношении агрегации тау-белка популяции клеток по сравнению с отрицательной в отношении агрегации тау-белка популяцией клеток, может указывать на то, что ген, на который нацелена данная гидовая РНК, проявляет синтетическую летальность с агрегатами тау-белка и является генетической уязвимостью, связанной с агрегацией тау-белка, или является потенциальной генетической уязвимостью, связанной с агрегацией тау-белка (например, кандидатом для дальнейшего тестирования с помощью вторичных скринингов), при этом ожидается, что нарушение гена, на который нацелена данная гидовая РНК, проявляет синтетическую летальность с агрегатами тау-белка. Паттерн более резкого истощения означает, что данная гидовая РНК истощается с большей скоростью с течением времени (например, различимый паттерн истощения при использовании дифференциального теста на вписанных в модель скоростях экспоненциального распада).
[00226] Подобным образом, описанные в данном документе линии биосенсорных клеток SAM/тау могут применяться в способах исключающего скрининга для выявления генетических уязвимостей, связанных с агрегацией тау-белка. Такие способы могут включать в себя обеспечение положительных в отношении агрегации тау-белка и отрицательных в отношении агрегации тау-белка популяций биосенсорных клеток SAM/тау, как описано в другом месте в данном документе, введение библиотеки, содержащей множество уникальных гидовых РНК, в каждую в популяцию, и определение содержания (например, числа считываний) каждой из указанного множества уникальных гидовых РНК во множестве моментов времени в исследовании с течением времени в каждой популяции клеток.
[00227] В качестве одного из примеров, способ может включать в себя обеспечение положительной в отношении агрегации тау-белка популяции клеток и отрицательной в отношении агрегации тау-белка популяции клеток, при этом каждая популяция клеток содержит химерный белок Cas, который содержит белок Cas с инактивированной нуклеазной активностью, слитый с одним или большим числом доменов активации транскрипции, химерный адаптерный белок, который содержит адаптерный белок, слитый с одним или большим числом доменов активации транскрипции, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, как описано в другом месте в данном документе. В положительной в отношении агрегации тау-белка популяции клеток первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, присутствуют стабильно в агрегированном состоянии, тогда как в отрицательной в отношении агрегации тау-белка популяции клеток первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, не присутствуют стабильно в агрегированном состоянии. Способ может далее включать в себя введение в каждую популяцию клеток библиотеки, содержащей множество уникальных гидовых РНК, которые нацелены на множество генов. Множество уникальных гидовых РНК образует комплексы с химерным белком Cas и химерным адаптерным белком, и данные комплексы активируют транскрипцию множества генов, что приводит к повышенной экспрессии генов. Наконец, содержание каждой из множества уникальных гидовых РНК может быть определено во множестве моментов времени в исследовании с течением времени в каждой популяции клеток. Истощение гидовой РНК в положительной в отношении агрегации тау-белка популяции клеток, но не в отрицательной в отношении агрегации тау-белка популяции клеток, может указывать на то, что ген, на который нацелена данная гидовая РНК, при активации его транскрипции проявляет синтетическую летальность с агрегатами тау-белка и является генетической уязвимостью, связанной с агрегацией тау-белка, или является потенциальной генетической уязвимостью, связанной с агрегацией тау-белка (например, кандидатом для дальнейшего тестирования с помощью вторичных скринингов), при этом ожидается, что активация транскрипции гена, на который нацелена данная гидовая РНК, проявляет синтетическую летальность с агрегатами тау-белка. В качестве альтернативы, паттерн более резкого истощения гидовой РНК с течением времени в положительной в отношении агрегации тау-белка популяции клеток по сравнению с отрицательной в отношении агрегации тау-белка популяцией клеток, может указывать на то, что ген, на который нацелена данная гидовая РНК, при активации его транскрипции проявляет синтетическую летальность с агрегатами тау-белка и является генетической уязвимостью, связанной с агрегацией тау-белка, или является потенциальной генетической уязвимостью, связанной с агрегацией тау-белка (например, кандидатом для дальнейшего тестирования с помощью вторичных скринингов), при этом ожидается, что активация транскрипции гена, на который нацелена данная гидовая РНК, проявляет синтетическую летальность с агрегатами тау-белка. Паттерн более резкого истощения означает, что данная гидовая РНК истощается с большей скоростью с течением времени (например, различимый паттерн истощения при использовании дифференциального теста на вписанных в модель скоростях экспоненциального распада).
[00228] Биосенсорные клетки Cas/тау, применяемые в указанном способе, могут представлять собой любые биосенсорные клетки Cas/тау, описанные в другой части данного документа. Аналогичным образом, биосенсорные клетки SAM/тау, применяемые в указанном способе, могут представлять собой любые биосенсорные клетки SAM/тау, описанные в другой части данного документа. Первый содержащий повторы домен тау-белка и второй содержащий повторы домен тау-белка могут быть разными, похожими или одинаковыми. Содержащий повторы домен тау-белка может представлять собой любой из содержащих повторы доменов тау-белка, описанных в другой части данного документа. Например, первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка могут представлять собой содержащие повторы домены тау-белка дикого типа, или могут содержать мутацию, способствующую агрегации, такую как мутация тау-белка P301S. Первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка могут содержать домен тау-белка с четырьмя повторами. В качестве одного конкретного примера, первый содержащий повторы домен тау-белка и (или) второй содержащий повторы домен тау-белка могут содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 11 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 11. В одном конкретном примере нуклеиновая кислота, кодирующая содержащий повторы домен тау-белка, может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 12 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 12, необязательно при этом данная нуклеиновая кислота кодирует белок, содержащий, состоящий по существу из, или состоящий из последовательности с SEQ ID NO: 11.
[00229] Первый содержащий повторы домен тау-белка может быть связан с первым репортером, а второй содержащий повторы домен тау-белка может быть связан со вторым репортером любыми способами. Например, репортер может быть слит с содержащим повторы доменом тау-белка (например, как часть слитого белка). Репортерные белки могут представлять собой любую пару репортерных белков, которые производят обнаруживаемый сигнал, когда первый содержащий повторы домен тау-белка, связанный с первым репортером, агрегирован со вторым содержащим повторы доменом тау-белка, связанным со вторым репортером. В качестве одного из примеров, первый и второй репортеры могут представлять собой расщепленный белок люциферазы. В качестве другого примера, первый и второй репортерные белки могут представлять собой пару резонансного переноса энергии флуоресценции (FRET-пару). FRET представляет собой физическое явление, при котором донорный флуорофор в возбужденном состоянии безызлучательно передает свою энергию возбуждения соседнему акцепторному флуорофору, тем самым заставляя акцептор испускать свою характерную флуоресценцию. Примеры FRET-nap (донорный и акцепторный флуорофоры) хорошо известны. См., например, работу Bajar et al. (2016) Sensors (Basel) 16 (9): 1488, которая включена в данный документ посредством ссылки во всей своей полноте для всех целей. В качестве конкретного примера FRET-пары, первый репортер может представлять собой голубой флуоресцентный белок (CFP), а второй репортер может представлять собой желтый флуоресцентный белок (YFP). В качестве конкретного примера, CFP может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 13 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 13. В качестве другого конкретного примера, YFP может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 15 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 15.
[00230] Для биосенсорных клеток Cas/тау белок Cas может представлять собой любой белок Cas, описанным в другом месте в данном документе. В качестве одного из примеров, белок Cas может представлять собой белок Cas9. Например, белок Cas9 представлять собой белок Cas9 из Streptococcus pyogenes. В качестве одного конкретного примера, белок Cas может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 21 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 21.
[00231] Один, или большее число, или все из следующего: белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, - могут стабильно экспрессироваться в популяции клеток. Например, нуклеиновые кислоты, кодирующие один, или большее число, или все из следующего: белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, могут быть геномно интегрированы в популяцию клеток. В одном конкретном примере нуклеиновая кислота, кодирующая белок Cas, может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 22 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 22, необязательно при этом данная нуклеиновая кислота кодирует белок, содержащий, состоящий по существу из, или состоящий из последовательности с SEQ ID NO: 21.
[00232] Для биосенсорных клеток SAM/тау белок Cas может представлять собой любой белок Cas, описанным в другом месте в данном документе. В качестве одного из примеров, белок Cas может представлять собой белок Cas9. Например, белок Cas9 представлять собой белок Cas9 из Streptococcus pyogenes. В качестве одного конкретного примера, химерный белок Cas может содержать белок Cas с инактивированной нуклеазной активностью, слитый с доменом активации транскрипции VP64. Например, химерный белок Cas может содержать, в направлении от N-конца до С-конца: белок Cas с инактивированной нуклеазной активностью; сигнал ядерной локализации; и домен активатора транскрипции VP64. В качестве одного конкретного примера, адаптерный белок может представлять собой белок оболочки MS2, и один или большее число доменов активации транскрипции в химерном адаптерном белке могут содержать домен активации транскрипции р65 и домен активации транскрипции HSF1. Например, химерный адаптерный белок может содержать, в направлении от N-конца до С-конца: белок оболочки MS2; сигнал ядерной локализации; домен активации транскрипции р65; и домен активации транскрипции HSF1. В одном конкретном примере нуклеиновая кислота, кодирующая химерный белок Cas, может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 38 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 38, необязательно при этом данная нуклеиновая кислота кодирует белок, содержащий, состоящий по существу из, или состоящий из последовательности с SEQ ID NO: 36. В одном конкретном примере нуклеиновая кислота, кодирующая химерный адаптерный белок, может содержать, состоять по существу из, или состоять из последовательности с SEQ ID NO: 39 или из последовательности, которая по меньшей мере на около 90%, на около 91%, на около 92%, на около 93%, на около 94%, на около 95%, на около 96%, на около 97%, на около 98% или на около 99% идентична последовательности с SEQ ID NO: 39, необязательно при этом данная нуклеиновая кислота кодирует белок, содержащий, состоящий по существу из, или состоящий из последовательности с SEQ ID NO: 37.
[00233] Один, или большее число, или все из следующего: химерный белок Cas, химерный адаптерный белок, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, - могут стабильно экспрессироваться в популяции клеток. Например, нуклеиновые кислоты, кодирующие один, или большее число, или все из следующего: химерный белок Cas, химерный адаптерный белок, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, - могут быть геномно интегрированы в популяцию клеток.
[00234] Как раскрыто в другом месте в данном документе, клетки могут представлять собой клетки любого типа. Например, клетки могут представлять собой эукариотические клетки, клетки млекопитающего или клетки человека (например, клетки НЕК293Т или нейрональные клетки).
[00235] Множество уникальных гидовых РНК можно ввести в указанные популяции клеток любыми известными способами. В некоторых способах гидовые РНК вводятся в указанные популяции клеток с помощью вирусной трансдукции, такой как ретровирусная, аденовирусная или лентивирусная трансдукция. В конкретном примере гидовые РНК могут вводятся с помощью лентивирусной трансдукции. Каждое из множества уникальных гидовых РНК может находиться в отдельном вирусном векторе. Указанные популяции клеток могут быть инфицированы с любым показателем множественности заражения. Например, показатель множественности заражения может составлять от около 0,1 до около 1,0, от около 0,1 до около 0,9, от около 0,1 до около 0,8, от около 0,1 до около 0,7, от около 0,1 до около 0,6, от около 0,1 до около 0,5, от около 0,1 до около 0,4 или от около 0,1 до около 0,3. В качестве альтернативы, показатель множественности заражения может составлять меньше чем около 1,0, меньше чем около 0,9, меньше чем около 0,8, меньше чем около 0,7, меньше чем около 0,6, меньше чем около 0,5, меньше чем около 0,4, меньше чем около 0,3 или меньше чем около 0,2. В конкретном примере показатель множественности заражения может составлять меньше чем около 0,3.
[00236] Гидовые РНК могут быть введены в указанные популяции клеток вместе с маркером отбора или репортерный геном для отбора клеток, которые имеют гидовые РНК, и способ может дополнительно включать в себя отбор клеток, которые содержат маркер отбора или репортерный ген. Примеры маркеров отбора и репортерных генов приведены в другой части данного документа. В качестве одного из примеров, маркер отбора может представлять собой маркер, который придает устойчивость к лекарственному средству, такой как неомицинфосфотрансфераза, гигромицин-В-фосфотрансфераза, пуромицин-N-ацетилтрансфераза и бластицидин-S-дезаминаза. Другой иллюстративный маркер отбора представляет собой белок устойчивости к блеомицину, кодируемый геном Sh ble (ген блеомицина из Streptoalloteichus hindustanus), который придает устойчивости к зеоцину (флеомицину D1). Например, клетки можно отбирать с помощью лекарственного средства (например, пуромицина), таким образом, что только клетки, трансдуцированные конструкцией гидовой РНК, сохраняются для дальнейшего применения в проведении скрининга. Например, лекарственное средство может представлять собой пуромицин или зеоцин (флеомицин D1).
[00237] В некоторых способах множество уникальных гидовых РНК вводят в концентрации, выбранной таким образом, чтобы большинство клеток получали только по одной из уникальных гидовых РНК. Например, если гидовые РНК вводят с помощью вирусной трансдукции, то клетки могут быть инфицированы с низким показателем множественности заражения, чтобы гарантировать, что большинство клеток с высокой вероятностью получат только одну вирусную конструкцию. В качестве одного конкретного примера, множественность заражения может составлять меньше чем около 0,3.
[00238] Указанные популяции клеток, в которые введено множество уникальных гидовых РНК, могут включать в себя любое подходящее число клеток. Например, указанные популяции клеток могут включать в себя больше чем около 50, больше чем около 100, больше чем около 200, больше чем около 300, больше чем около 400, больше чем около 500, больше чем около 600, больше чем около 700, больше чем около 800, больше чем около 900, или больше чем около 1000 клеток на уникальную гидовую РНК. В конкретном примере указанные популяции клеток включают в себя больше чем около 300 клеток, или больше чем около 500 клеток на уникальную гидовую РНК.
[00239] Множество уникальных гидовых РНК может быть нацелено на любое число генов. Например, множество уникальных гидовых РНК может быть нацелено на около 50 или большее число генов, на около 100 или большее число генов, на около 200 или большее число генов, на около 300 или большее число генов, на около 400 или большее число генов, на около 500 или большее число генов, на около 1000 или большее число генов, на около 2000 или большее число генов, на около 3000 или большее число генов, на около 4000 или большее число генов, на около 5000 или большее число генов, на около 10000 или большее число генов, или на около 20000 или большее число генов. В некоторых способах гидовые РНК могут быть выбраны для нацеливания на гены в конкретном сигнальном пути. В некоторых способах библиотека уникальных гидовых РНК представляет собой полногеномную библиотеку.
[00240] Множество уникальных гидовых РНК может быть нацелено на любое число последовательностей в каждом отдельном целевом гене. В некоторых способах, нацеливание происходит в среднем на множество целевых последовательностей в каждом из целевого множества генов. Например, нацеливание происходит в среднем на от около 2 до около 10, от около 2 до около 9, от около 2 до около 8, от около 2 до около 7, от около 2 до около 6, от около 2 до около 5, от около 2 до около 4 или от около 2 до около 3 уникальных целевых последовательностей в каждом из целевого множества генов. Например, нацеливание происходит в среднем на по меньшей мере около 2, на по меньшей мере около 3, на по меньшей мере около 4, на по меньшей мере около 5 или на по меньшей мере около 6 уникальных целевых последовательностей в каждом из целевого множества генов. В качестве конкретного примера, нацеливание происходит в среднем на около 6 целевых последовательностей в каждом из целевого множества генов. В качестве другого конкретного примера, нацеливание происходит в среднем на от около 3 до около 6 или на от около 4 до около 6 целевых последовательностей в каждом из целевого множества генов.
[00241] Гидовые РНК могут быть нацелены на любую желаемую область в целевых генах. В некоторых способах CRISPRn, в которых применяются биосенсорные клетки Cas/тау, каждая гидовая РНК нацелена на конститутивный экзон, если это возможно. В некоторых способах каждая гидовая РНК нацелена на конститутивный 5'-экзон, если это возможно. В некоторых способах каждая гидовая РНК нацелена на первый экзон, второй экзон или третий экзон (с 5'-конца гена), если это возможно. В некоторых способах CRISPRa, в которых применяются биосенсорные клетки SAM/тау, каждая гидовая РНК может быть нацелена на целевую последовательность гидовой РНК в пределах 200 п. о. в направлении к 5' от сайта начала транскрипции, если это возможно. В некоторых способах CRISPRa, в которых применяются биосенсорные клетки SAM/тау, каждая гидовая РНК может содержать один или большее число адаптер-связывающих элементов, с которыми может специфически связываться химерный адаптерный белок. В одном из примеров каждая гидовая РНК содержит два адаптер-связывающих элемента, с которыми может специфически связываться химерный адаптерный белок, необязательно при этом первый адаптер-связывающий элемент находится внутри первой петли каждой из одной или большего числа гидовых РНК, а второй адаптер-связывающий элемент находится внутри второй петли каждой из одной или большего числа гидовых РНК. Например, адаптер-связывающий элемент может содержать последовательность, указанную в SEQ ID NO: 33. В конкретном примере каждая из одной или большего числа гидовых РНК представляет собой единую гидовую РНК, содержащую часть CRISPR-PHK (крРНК), слитую с частью трансактивирующей CRISPR-PHK (тракрРНК), и первая петля представляет собой тетрапетлю, соответствующую остаткам 13-16 из последовательности с SEQ ID NO: 17, 19, 30 или 31, а вторая петля представляет собой шпильку 2, соответствующую остаткам 53 - 56 из последовательности с SEQ ID NO: 17, 19, 30 или 31.
[00242] Содержание гидовых РНК может быть определено любыми подходящими способами. В конкретном примере содержание определяется с помощью секвенирования нового поколения. Секвенирование нового поколения относится к технологиям высокопроизводительного секвенирования ДНК, не основанным на методе Сэнгера. Например, определение содержания гидовой РНК может включать в себя измерение числа считываний гидовой РНК.
[00243] Указанное исследование с течением времени на стадии (с) может иметь любую подходящую продолжительность. Например, указанное исследование с течением времени может иметь продолжительность по меньшей мере около 1-й недели, по меньшей мере около 2-х недель, больше чем около 1-й недели или больше чем около 2-х недель. Например, указанное исследование с течением времени может иметь продолжительность от около 6 суток до около 28 суток, от около 9 суток до около 25 суток, от около 12 суток до около 22 суток, от около 15 суток до около 19 суток, или около 17 суток. Подобным образом, указанное исследование с течением времени на стадии (с) может включать в себя любое подходящее число пассажей клеток или удвоений клеток. Указанное исследование с течением времени может включать в себя от около 5 до около 20, от около 6 до около 19, от около 7 до около 18, от около 8 до около 17, от около 9 до около 16, от около 10 до около 15, от около 11 до около 14, от около 12 до около 13 или около 12 удвоений клеток. В качестве альтернативы, указанное исследование с течением времени может включать в себя от около 2 до около 8, от около 2 до около 7, от около 2 до около 6, от около 3 до около 5, от около 3 до около 6, от около 3 до около 7, от около 3 до около 8 или около 4 пассажей клеток. Например, может происходить от 10 до 15 удвоений клеток. В конкретном примере между первым сбором в 3-й сутки и 17-ми сутками может произойти около 12 удвоений клеток (т.е. около 4 пассажей клеток). В некоторых из способов для поддержания хорошей репрезентации полногеномной библиотеки гРНК клетки не разбавляют слишком сильно при каждом пассаже. Например, клетки могут быть пассированы как около 1/10, около 1/9, около 1/8, около 1/7, около 1/6, около 1/5, около 1/4, около 1/3 или около 1/2. В конкретном примере клетки пассированы на около 1/5 (например, так, чтобы было ≥2 удвоения клеток).
[00244] Описанные в данном документе клетки-биосенсоры тау-белка праймированы для агрегации. Это значит, что они находятся на переломном этапе агрегации. Мы обнаружили, что агрегаты тау-белка вызывают сильное и последовательное изменение профиля транскрипции клеток-биосенсоров тау-белка. Мы предположили, что эти положительные в отношении агрегации клетки могут быть избирательно уязвимы к определенным генетическим нарушениям, в отличие от отрицательных в отношении агрегации клеток. Мы также предположили, что любая «мутация» при скрининге CRISPRn, которая может вызвать «специфическую» гибель клеток, содержащих агрегаты, также может иметь меньший вредный эффект на клетки, не содержащие агрегаты. Аналогичным образом, любая активация гена при скрининге CRISPRa, которая может вызвать «специфическую» гибель клеток, содержащих агрегаты, также может иметь меньший вредный эффект на клетки, не содержащие агрегаты. Оценка во множестве моментов времени позволяет нам идентифицировать цель данного типа, определяя профили истощения РНК (скорость истощения или резкость истощения гРНК) с течением времени, а не только при последнем пассаже по сравнению с первым пассажем, ведь при последнем подходе цель данного типа будет пропущена. Например, данный тип профиля истощения наблюдался с целевым геном 1. Мы идентифицировали данный тип профилей истощения для нашей ведущей цели -целевого гена 1.
[00245] Указанное множество моментов времени на стадии (с) может представлять собой любое подходящее число моментов времени. Например, указанное множество моментов времени на стадии (с) может составлять по меньшей мере около 2 моментов времени, по меньшей мере около 3 моментов времени, по меньшей мере около 4 моментов времени, по меньшей мере около 5 моментов времени, по меньшей мере около 6 моментов времени, больше чем около 2 моментов времени, больше чем около 3 моментов времени, больше чем около 4 временных точек, больше чем около 5 моментов времени или больше чем около 6 моментов времени. Например, может быть от около 3 до около 9 моментов времени, от около 4 до около 8 моментов времени, от около 5 до около 7 моментов времени или около 6 моментов времени. В качестве другого примера, может быть от около 2 до около 6 моментов времени, от около 3 до около 5 моментов времени или около 4 моментов времени.
[00246] Каждый момент времени может соответствовать пассажу клеток. Промежуток времени между каждым последующим моментом времени может быть равным любому подходящему периоду времени. Например, между каждым моментом времени может проходить по меньшей мере около 12 часов, по меньшей мере около 1 суток, по меньшей мере около 2 суток, по меньшей мере около 3 суток, больше чем около 12 часов, больше чем около 1 суток, больше чем около 2 суток или больше чем около 3 суток. Например, между каждым последующим моментом времен может проходить от около 1 суток до около 6 суток, от около 2 суток до около 5 суток или от около 3 суток до около 4 суток. В качестве конкретного примера, между каждым последующим моментом времени (т.е. между последовательными пассажами клеток) может проходить по меньшей мере около 2 суток или от около 3 суток до около 4 суток.
[00247] Первый момент времени может представлять собой любой подходящий момент времени после введения гидовых РНК в указанные популяции клеток. Например, для способов скрининга CRISPRn с использованием биосенсорных клеток Cas/тау, первый момент времени может наступить по прошествии такого количества времени, которое достаточно для того, чтобы гидовые РНК образовали комплексы с белком Cas, и белок Cas расщепил множество генов, приведя к нокауту генной функции. Аналогичным образом, для способов скрининга CRISPRa с использованием биосенсорных клеток SAM/тау, первый момент времени может наступить по прошествии такого количества времени, которое достаточно для того, чтобы гидовые РНК образовали комплексы с химерным белком Cas и химерным адаптерным белком, и чтобы данные комплексы активировали транскрипцию множества генов, приведя к повышенной экспрессии генов. Например, первый момент времени может наступить спустя по меньшей мере около 1 суток, спустя по меньшей мере около 2 суток, спустя по меньшей мере около 3 суток, спустя больше чем около 1 суток, спустя больше чем около 2 суток или спустя больше чем около 3 суток после введения гидовых РНК в клетки. В качестве альтернативы, первый момент времени может наступить спустя от около 0 суток до около 6 суток, спустя от около 1 суток до около 5 суток, спустя от около 2 суток до около 4 суток, или спустя около 3 суток после введения гидовых РНК в клетки.
[00248] В одном конкретном примере содержание гидовых РНК оценивают на 3-й, 6-е или 7-е сутки, на 10-е или 11-е сутки, на 13-е или 14-е сутки и на 17-е сутки после введения гидовых РНК в 0-е сутки. Например, содержание гидовых РНК можно оценивать на 3-й, 7-е, 10-е, 14-е и 17-е сутки после введения гидовых РНК в 0-е сутки.
[00249] Определение того, является гидовая РНК истощенной с течением времени или нет, может включать в себя разные оценки. В качестве одного из примеров, гидовая РНК может считаться истощенной, если содержание данной гидовой РНК в каждый из моментов времени является меньшим или равным содержанию данной гидовой РНК в предшествующий момент времени. В качестве другого примера, гидовая РНК может считаться истощенной на стадии (с), если содержание данной гидовой РНК в каждый из моментов времени после второго момента времени является меньшим или равным содержанию в момент времени, на два момента времени предшествующий данному моменту времени (т.е. в момент времени перед предшествующим моментом времени, такой как момент времени 3 по сравнению с моментом времени 1, момент времени 4 по сравнению с моментом времени 2, момент времени 5 по сравнению с моментом времени 3 и т.д.). В одном конкретном примере содержание гидовых РНК оценивают на 3-й, 6-е или 7-е сутки, на 10-е или 11-е сутки, на 13-е или 14-е сутки и на 17-е сутки после введения гидовых РНК в 0-е сутки, и гидовая РНК считается истощенной, если ее содержание в 10-е или 11-е сутки является меньшим, чем в 3-й сутки, ее содержание в 13-е или 14-е сутки является меньшим, чем в 6-е или 7-е сутки, и ее содержание в 17-е сутки является меньшим, чем в 10-е или 11-е сутки.
[00250] Оценка в описанных в данном документе способах может также учитывать тенденцию среди всех гидовых РНК в библиотеке, нацеленных на конкретный ген. Например, можно считать, что ген проявляет синтетическую летальность с агрегатами тау-белка, если по меньшей мере около 30%, по меньшей мере около 33%, по меньшей мере около 35%, по меньшей мере около 40%, по меньшей мере около 45%, по меньшей мере около 50%, по меньшей мере около 60%, по меньшей мере около 65%, по меньшей мере около 70%, по меньшей мере около 75%, больше чем около 30%, больше чем около 33%, больше чем около 35%, больше чем около 40%, больше чем около 45%, больше чем около 50%, больше чем около 60%, больше чем около 65%, больше чем около 70% или больше чем около 75% (например, по меньшей мере около 30% или больше чем около 30%) гидовых РНК в данной библиотеке, нацеленных на ген, истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток. В качестве некоторых конкретных неограничивающих примеров, можно считать, что ген проявляет синтетическую летальность с агрегатами тау-белка (или ожидается, что ген будет проявлять синтетическую летальность с агрегатами тау-белка) в следующих ситуациях: (1) если в библиотеке есть одна гидовая РНК, которая нацелена на данный ген, и данная одна гидовая РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (2) если в библиотеке есть две гидовые РНК, которые нацелены на данный ген, и по меньшей мере одна из данных двух гидовых РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (3) если в библиотеке есть три гидовые РНК, которые нацелены на данный ген, и по меньшей мере одна из данных трех гидовых РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (4) если в библиотеке есть четыре гидовые РНК, которые нацелены на данный ген, и по меньшей мере две из данных четырех гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; (5) если в библиотеке есть пять гидовых РНК, которые нацелены на данный ген, и по меньшей мере две из данных пяти гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; и (6) если в библиотеке есть шесть гидовых РНК, которые нацелены на данный ген, и по меньшей мере три из данных шести гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток.
[00251] Оценка в описанных в данном документе способах может также учитывать тенденцию гидовых РНК в библиотеке, нацеленных на конкретный ген, в ходе множества экспериментов. Например, способ можно повторить по меньшей мере около 2 раз, по меньшей мере около 3 раз, по меньшей мере около 4 раз, по меньшей мере около 5 раз, по меньшей мере около 6 раз, больше чем около 1 раза, больше чем около 2 раз, больше чем около 3 раз, больше чем около 4 раз, больше чем около 5 раз или больше чем около 6 раз. Например, можно считать, что ген проявляет синтетическую летальность с агрегатами тау-белка, если он выбирается (т.е. если считается, что он проявляет синтетическую летальность с агрегатами тау-белка) в по меньшей мере около 30%, в по меньшей мере около 35%, в по меньшей мере около 40%, в по меньшей мере около 45%, в по меньшей мере около 50%, в по меньшей мере около 55%, в по меньшей мере около 60%, в по меньшей мере около 65%, в по меньшей мере около 70%, в по меньшей мере около 75%, в больше чем около 30%, в больше чем около 35%, в больше чем около 40%, в больше чем около 45%, в больше чем около 50%, в больше чем около 55%, в больше чем около 60%, в больше чем около 65%, в больше чем около 70% или в больше чем около 75% всех экспериментов. Например, можно считать, что ген проявляет синтетическую летальность с агрегатами тау-белка, если он выбирается (т.е. если считается, что он проявляет синтетическую летальность с агрегатами тау-белка) в больше чем около 50% или в больше чем около 60% всех экспериментов. В качестве конкретного примера, указанные способы можно повторить в трех экспериментах, и можно считать, что ген проявляет синтетическую летальность с агрегатами тау-белка (или может быть выбран в качестве кандидата для проявления синтетической летальности с агрегатами тау-белка (например, для дальнейшего тестирования во вторичных экранах)), если выбирается (т.е. если считается, что он проявляет синтетическую летальность с агрегатами тау-белка) в по меньшей мере двух из трех экспериментов.
[00252] Все патентные документы, веб-сайты, другие публикации, учетные номера и т.п., указанные выше или ниже в данном документе, включены в данный документ посредством ссылки в полном объеме для всех целей в той же степени, как если бы каждый отдельный элемент был специально и отдельно указан как включенный в данный документ посредством ссылки. Если разные версии последовательности связаны с учетным номером в разное время, то подразумевается та версия, которая связана с учетным номером на действительную дату подачи данной заявки. Действительная дата подачи означает более раннюю дату из даты фактической подачи или даты подачи приоритетной заявки со ссылкой на учетный номер, если это применимо. Аналогичным образом, если разные версии публикации, веб-сайта и т.п. публикуются в разное время, то подразумевается та версия, которая была опубликована последней на действительную дату подачи данной заявки, если не указано иное. Любые признак, стадия, элемент, вариант осуществления или аспект данного изобретения можно применять в комбинации с любым другим, если специально не указано иное. Несмотря на то, что данное изобретение описано довольно подробно с помощью иллюстраций и примеров для ясности и понимания, будет очевидно, что могут быть осуществлены определенные изменения и модификации в пределах объема прилагаемой формулы данного изобретения.
КРАТКОЕ ОПИСАНИЕ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
[00253] Нуклеотидные и аминокислотные последовательности, перечисленные в прилагаемом перечне последовательностей, показаны с использованием стандартных буквенных сокращений для нуклеотидных оснований и трехбуквенного кода для аминокислот.Нуклеотидные последовательности следуют стандартному соглашению: они начинаются с 5'-конца последовательности и простираются вперед (т.е. слева направо в каждой строке) до 3'-конца. Показана только одна цепь каждой нуклеотидной последовательности, но подразумевается, что комплементарная ей цепь включена посредством любой ссылки на отображаемую цепь. Когда представлена нуклеотидная последовательность, кодирующая аминокислотную последовательность, следует понимать, что также представлены ее варианты с вырожденными ко донами, которые кодируют ту же аминокислотную последовательность. Аминокислотные последовательности следуют стандартному соглашению: они начинаются с амино-конца последовательности и простираются вперед (т.е. слева направо в каждой строке) до карбокси-конца.
ПРИМЕРЫ
Пример 1. Разработка полногеномной скрининговой платформы CRISPR/Cas9 для идентификации генетических уязвимостей, связанных с агрегацией тау-белка
[00255] Аномальная агрегация или фибриллизация белков является определяющим признаком многих заболеваний, в частности, ряда нейродегенеративных заболеваний, таких как болезнь Альцгеймера (БА), болезнь Паркинсона (БП), лобно-височная деменция (ЛВД), боковой амиотрофический склероз (БАС), хроническая травматическая энцефалопатия (ХТЭ), болезнь Крейтцфельда Якоба (БКЯ) и другие. Во многих из данных заболеваний фибриллизация определенных белков в нерастворимые агрегаты является не только характерным признаком заболевания, но также считается причинным фактором нейротоксичности. Кроме того, данные заболевания характеризуются распространением совокупной патологии через центральную нервную систему в соответствии со стереотипными паттернами - процессом, который коррелирует с прогрессированием заболевания. Таким образом, идентификация генов и генетических путей, которые модифицируют процессы аномальной агрегации белков или распространения агрегатов от клетки к клетке, имеет большое значение для лучшего понимания этиологии нейродегенеративных заболеваний, а также для разработки стратегий терапевтического вмешательства.
[00256] Чтобы идентифицировать гены и пути, которые проявляют синтетическую летальность с ассоциированными с заболеванием белковыми агрегатами, была разработана платформа для проведения полногеномных скринингов с использованием библиотек егРНК нуклеаз CRISPR (CRISPRn). Указанная платформа идентифицирует гены, нарушение которых вызывает гибель клеток, особенно в контексте аномальной агрегации белка. Идентификация таких генов может пролить свет на механизмы нейротоксичности, связанной с агрегатами, и генетические пути, которые способствуют гибели нейронов в контексте нейродегенеративного заболевания.
[00257] В данном скрининге использовалась линия клеток-биосенсоров тау-белка человека, состоящая из клеток НЕК293Т, стабильно экспрессирующих домен тау-белка с четырьмя повторами, tau_4RD, содержащий домен связывания микротрубочек (MBD) тау-белка с патогенной мутацией P301S, слитый либо с CFP, либо с YFP. Это значит, что указанные клеточные линии НЕК293Т содержат два трансгена, стабильно экспрессирующих варианты ассоциированного с заболеванием белка, слитые с флуоресцентным белком CFP или с флуоресцентным белком YFP - тау4RD-CFP/тау4RD-YFP (TCY), где содержащий повторы домен тау-белка (4RD) содержит патогенную мутацию P301S. См. фиг. 1. В данных биосенсорных линиях агрегация белков тау-CFP/тау-YFP производит FRET-сигнал, который является результатом переноса энергии флуоресценции от донора CFP к акцептору YFP. См. фиг. 2. Клетки, положительные в отношении FRET, содержащие агрегаты тау-белка, могут быть отсортированы и выделены с помощью метода проточной цитометрии. В исходном состоянии нестимулированные клетки экспрессируют репортеры в стабильном растворимом виде с минимальным сигналом FRET. При стимуляции (например, липосомная трансфекция засеваемыми частицами) репортерные белки образуют агрегаты, производя сигнал FRET. Клетки, содержащие агрегаты, можно выделить с помощью метода FACS. Стабильно размножающиеся клеточные линии, содержащие агрегаты, Аг[+], могут быть выделены путем клонального серийного разведения клеточных линий Аг[-].
[00258] В данную линию клеток-биосенсоров тау-белка было внесено несколько модификаций, чтобы сделать ее пригодной для генетического скрининга. Сначала данные клетки-биосенсоры тау-белка были модифицированы путем введения Cas 9-экспремирующего трансгена с помощью лентивирусного вектора. Клональные трансгенные клеточные линии, экспрессирующие Cas9, были отобраны с помощью бластицидина и выделены путем клонального серийного разведения для получения клонов, происходящих из одиночных клеток. В данных клонах проводили оценку уровня экспрессии Cas9 с помощью кПЦР-РВ (фиг. 3А) и активности расщепления ДНК с помощью цифровой ПЦР (фиг. 3В). Относительные уровни экспрессии Cas9 также показаны в таблице 3.
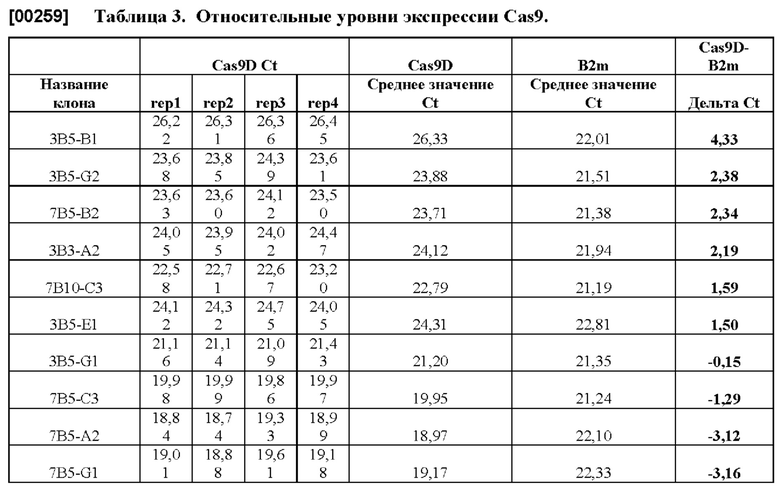
[00260] В частности, эффективность мутации Cas9 оценивали с помощью цифровой ПЦР через 3 суток и через 7 суток после трансдукции лентивирусов, кодирующих гРНК против двух выбранных целевых генов. Эффективность разрезания ограничивалась уровнями Cas9 в клонах с более низкой экспрессией. Клон с достаточным уровнем экспрессии Cas9 был необходим для достижения максимальной активности. Несколько из клонов-производных с более низкой экспрессией Cas9 не смогли эффективно разрезать целевые последовательности, тогда как клоны с более высокой экспрессией (включая те, которые использовались для скрининга) были способны генерировать мутации в целевых последовательностях в генах PERK и SNCA с эффективностью около 80% после трех суток в культуре. Эффективное разрезание наблюдали уже через 3 суток после трансдукции гРНК с лишь незначительным улучшением через 7 суток. Клон 7В10-С3 был выбран как высокопроизводительный клон для применения в последующих скринингах библиотек.
[00261] Затем были получены субклоны этих Cas9-экспрессирующих биосенсорных линий тау-CFP/тау-YFP (TCY), в которых тау-белок стабильно присутствует либо в неагрегированном состоянии (состояние по умолчанию) (Аг[-]), либо в агрегированном состоянии (Аг[+]). Для получения клеточных линий, в которых тау-белок стабильно и постоянно присутствует в агрегированном состоянии, клетки, экспрессирующие Cas9, обрабатывали рекомбинантным фибриллизованным тау-белком, смешанным с липофектаминовым реагентом, чтобы «засеять» агрегацию тау-белка, трансгенно экспрессируемого данными клетками. См. фиг. 5. Затем «засеянные» клетки серийно разбавляли для получения клонов, полученных из отдельных клеток, и данные клоны затем размножали для идентификации клональных клеточных линий, в которых агрегаты тау-белка стабильно сохраняются во всех клетках при росте и множественных пассажах с течением времени.
[00262] Затем мы проанализировали субклоны Аг[+] и Аг[-], чтобы определить, были ли определенные гены нарушены агрегацией тау-белка. См. фиг. 6. Неконтролируемая кластеризация показала, что субклоны Аг[+] и субклоны Аг[-] различались по статусу агрегации (процедура кластеризации: логарифмическая трансформация считываний на миллион т.п.и. (RPKM); метрика расстояния: 1 абсолютное значение коэффициента корреляции Пирсона; иерархическая кластеризация). См. фиг. 7. Неконтролируемая кластеризация относится к кластеризации образцов без каких-либо предварительных знаний о данных образцах (например, из какой линии клеток взят образец (Аг[+] или Аг[-])), так что формирование кластера среди образцов полностью определяется самими данными. Процедура была следующей: (1) прибавление 1 к уровню экспрессии всех генов (единица представляет собой число считываний на миллион т.п.и (RPKM) в каждом образце; (2) log2-трансформация значений RPKM+1; (3) вычисление попарного коэффициента корреляции Пирсона среди всех образцов; (4) вычисление сходства между любой парой образцов как 1 абсолютного значения коэффициента корреляции Пирсона; и (5) применение стандартного алгоритма иерархической кластеризации к значениям из п. (4). Полученные данные показали, что образцы клеточных линий Аг[+] и Аг[-] образуют четко разделенные кластеры исключительно на основе профилей экспрессии их генов, что позволяет предположить, что линии клеток Аг[+] и Аг[-] различаются на уровне экспрессии генов.
[00263] РНК-секвенирование субклонов Аг[+] и Аг[-] биосенсора тау-белка показало, что более 1500 генов нарушены агрегацией тау-белка. См. фиг. 8. Значительно нарушенные гены определялись как гены, у которых кратность изменения ≥1,5 в любом направлении и значение р≤0,01. РНК-секвенирование пяти субклонов Аг[+] и пяти субклонов Аг[-] показало, что присутствие агрегатов тау-белка вызывает сильное и последовательное изменение профиля транскрипции суб клонов Аг[+] по сравнению с субклонами Аг[-]. Таким образом, субклоны Аг[+] могут быть избирательно уязвимы к определенным генетическим нарушениям, в отличие от субклонов Аг[-]. Как объясняется в примере 2, мы исследовали эти уязвимости с помощью библиотек CRISPR с целью выполнения исключающего скрининга и идентификации тех егРНК, которые вызывают синтетическую летальность в сочетании с агрегатами тау-белка.
Пример 2. Полногеномный скрининг CRISPR/Cas9 для идентификации генетических уязвимостей, связанных с агрегацией тау-белка
[00264] Чтобы исследовать уязвимости, идентифицированные в примере 1, был проведен объединенный полногеномный скрининг нуклеаз CRISPR (CRISPRn) для выявления генетических уязвимостей, связанных с агрегацией тау-белка. Более конкретно, биосенсорные клетки тау-СFР/тау-YFP, экспрессирующие Cas9 и либо содержащие агрегаты (Аг[+]), либо не содержащие агрегаты (Аг[-]), трансдуцировали двумя полногеномными библиотеками CRISPR егРНК человека с использованием подхода лентивирусной доставки для введения нокаутирующих мутаций в каждый целевой ген в количестве 500Х клеток на егРНК. См. фиг. 4 и фиг. 9. Библиотеки CRISPR егРНК нацелены на 5'-конститутивные экзоны для функционального нокаута со средним охватом 3-4 егРНК на ген. Указанные егРНК были разработаны таким образом, чтобы избежать нецелевых эффектов, избегая егРНК с двумя или меньшим числом несовпадений с нецелевыми геномными последовательностями. Указанные библиотеки охватывают 19050 генов человека и 1864 микроРНК с 1000 контрольными ненацеленными егРНК. Указанные библиотеки трансдуцировали с показателем множественности заражения (МЗ, англ. «MOI»)<0,3 при охвате >500 клеток на егРНК. Клетки-биосенсоры тау-белка выращивали в условиях отбора пуромицином для отбора клеток с интеграцией и экспрессией уникальной егРНК на клетку. Отбор пуромицином начинали через 24 часа после трансдукции при концентрации, составляющей 1 мкг/мл.
[00265] Образцы клеток (Аг[+]) и (Аг[-]) собирали в пяти моментах времени: 3-й сутки, 6-е - 7-е сутки, 10-е - 11-е сутки, 13-е - 14-е сутки и 17-е сутки после трансдукции. Выделение ДНК и ПЦР-амплификация интегрированных конструкций егРНК позволили провести характеризацию репертуара егРНК в каждой линии клеток в каждый момент времени с помощью секвенирования нового поколения (СНП). Данный скрининг состоял из 3-х повторных экспериментов. Анализ данных СНП позволил идентифицировать основные гены, состоящие из тех генов, чьи нацеленные егРНК со временем истощаются как в линиях клеток Аг[+], так и в линиях клеток Аг[-]. См. фиг. 10А. Что еще более важно, данный способ скрининга позволил идентифицировать синтетически летальные гены, состоящие из генов, чьи нацеленные егРНК со временем истощаются преимущественно в линиях клеток Аг[+] по сравнению с линиями клеток Аг[-]. См. фиг. 10 В.
[00266] Профили истощения оценивались с использованием недавно определенного анализа динамики изменений во времени, в котором егРНК с паттерном последовательного снижения СНП-считываний от наиболее раннего момента времени (3-й сутки) до конечного момента времени (17-е сутки) считались истощенными. См. фиг. 11 и таблицу 4.

[00268] Это новый аналитический подход к оценке истощения егРНК по сравнению с более традиционным подходом, заключающимся в простом сравнении СНП-считываний коллекции конечных клеток с первым пассажем. Для сравнения, другие опубликованные полногеномные скрининги CRISPRn на предмет истощения сравнивали показатели CRISPR как отношение числа считываний между финальным сбором и ранним пассажем. Напротив, наш подход состоял в том, чтобы изучить полную динамику во времени с целью идентификации генов, которые проявляют паттерн истощения егРНК с течением времени. Анализ динамики во времени выявил больше генов егРНК, истощенных в субпопуляции Аг[+], чем в субпопуляции Аг[-] во всех трех повторных экспериментах.
[00269] Первые стадии данного анализа включали в себя контроль качества (ненацеленные егРНК), нормализацию (относительно общего числа считываний на образец) и отметку «присутствия»/«отсутствия» для каждой егРНК на образец с порогом обнаружения, равным 30 считываний. Сначала исследовали ненацеленные егРНК. Было подтверждено отсутствие сильного или систематического нарушения или истощения 1000 ненацеленных контрольных егРНК. Затем были проведены эксперименты с целью демонстрации того, что плазмидная библиотека егРНК и образцы на 3-й сутки были очень похожи в отношении числа считываний егРНК (на 3-й сутки не произошло значительного истощения егРНК). Это было подтверждением для использования 3-х суток в качестве контрольной точки для анализа динамики с течением времени.
[00270] Истощенные гены затем идентифицировали с помощью анализа динамики с течением времени. В каждом повторе отбирали истощенные егРНК с невозрастающими временными паттернами (число считываний на 10-е сутки ≤ на 3-й сутки; число считываний на 14-е сутки ≤ на 6-е сутки; число считываний на 17-е сутки ≤ на 10-е сутки) относительно 3-х суток. Сохраняли егРНК, уже истощенные на 3-й сутки (т.е. те, которые были ниже предела обнаружения, равного 30, на 3-й сутки, и которые оставались ниже предела обнаружения в остальные моменты времени). Флуктуации, близкие к пределу обнаружения, игнорировали (т.е. если число считываний для данной егРНК было меньшим или равным среднему числу считываний егРНК, которые были ниже предела обнаружения+2 * стандартное отклонение числа считываний егРНК, которые были ниже предела обнаружения, то такую егРНК считали необнаруженной). Гены отбирали, если для них имелось достаточное число истощенных егРНК (1 из 1 суммарной егРНК, 1 из 2 суммарных егРНК, 1 из 3 суммарных егРНК, 2 из 4 суммарных егРНК, 2 из 5 суммарных егРНК или 3 из 6 суммарных егРНК). Гены сохраняли, если они отбирались по крайней мере в 2 из 3 повторных экспериментов по исследованию динамики с течением времени. Затем гены идентифицировали как «основные», если егРНК, нацеленные на данные гены, были истощены как в Аг[+], так и в Аг[-]. Гены идентифицировали как «синтетически летальные», если егРНК, нацеленные на данные гены, были истощены в Аг[+], но не в Аг[-] (отсутствие истощения в Аг[-] по сравнению с Аг[+]; отсеивание на основании нескольких повторов и ручной проверки). Под отсеиванием подразумевается сбор генов, представленных во всех трех повторных экспериментах по исследованию динамики с течением времени в клеточной линии Аг[+], а затем исключение любого гена, представленного по меньшей мере в одном эксперименте в клеточной линии Аг[-]. Ручная проверка включала в себя просмотр гидовых РНК, истощенных в одном из экспериментов с Аг[+], поиск гидовых РНК, истощенных во всех экспериментах с Аг[+], но ни в одном из экспериментов с Аг[-] или только в одном из экспериментов с Аг[-], и подтверждение того, что ни одна цель не была пропущена.
[00271] Сильное или систематическое нарушение с 1000 контрольных ненацеленных егРНК отсутствовало (данные не показаны). В частности, для каждого из 6-х, 7-х, 10-х, 11-х, 13-х, 14-х и 17-х суток по сравнению с 3-й сутками все экспериментальные повторы были объединены как для Аг[+], так и для Аг[-]. Рассчитывали р-значения (скорректированные для множественного тестирования) изменений в числе считываний. Ни одна из гРНК в любой момент времени не имела значения р<0,05.
[00272] Анализ динамики с течением времени показал, что 977 генов являются «основными» генами с егРНК, имеющими паттерны истощения во времени как в субклонах Аг[+], так и в субклонах Аг[-] (данные не показаны). Данные гены, определенные как «основные», значительным образом перекрываются с генами, которые определены как основные в общедоступных наборах данных (см., например, работу Tzelepis et al. (2016) Cell Reports 17: 1193 - 1205 и работу Wang et al. (2015) Science 350 (6264): 1096 - 1101, каждая из которых включена в данный документ посредством ссылки во всей своей полноте и для всех целей). См. фиг. 13 и таблицу 5.
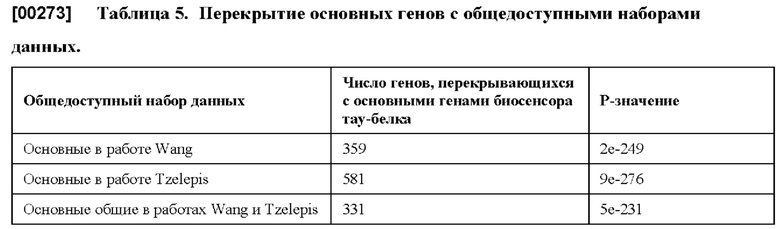
[00274] Гены, на которые нацелены множественные егРНК, проявляющие паттерн истощения при множественных повторных скринингах в Аг[+], но не в Аг[-], были отобраны для дальнейшей валидации во вторичном скрининге как «синтетически летальные» гены. Семьдесят один ген был идентифицирован как истощенный во всех экспериментальных повторностях для Аг[+] по сравнению с 3-й сутками, но не в одной из экспериментальных повторностей для Аг[-]. См. фиг. 14. Данные для трех целевых генов (целевой ген 1, целевой ген 5 и целевой ген 6) представлены на фиг. 15 В.
[00275] Данный 71 ген был идентифицирован путем анализа истощения егРНК в трех повторах, истощения егРНК в нескольких моментах времени и путем визуальной проверки данных. Затем были созданы специальные мини-библиотеки GenScript. Библиотека 1 содержала 462 уникальные нацеленные егРНК (нацеленные на 71 предполагаемый синтетически летальный ген с егРНК, идентифицированными как избирательно истощенные в клетках Аг[+], и на 10 предполагаемых основных генов с егРНК, идентифицированными как истощенные в клетках Аг[+] и Аг[-], на каждый нацелено по ~ 6 уникальных егРНК). Библиотека 2 содержала 500 уникальных ненацеленных контрольных егРНК, которые не изменялись в течение исследования динамики со временем. Распределение числа считываний для двух указанных библиотек показано на фиг. 16 и фиг. 17, соответственно. Это показывает, что репрезентация двух указанных библиотек сохраняется после размножения. Коэффициент г Пирсона описывает статистическую корреляцию между исходной библиотекой и библиотекой после размножения.
[00276] Затем указанные библиотеки использовались для вторичного скрининга потенциальных целей. См. фиг. 18. Истощение нацеленных егРНК по сравнению с ненацеленными контрольными егРНК оценивали на протяжении 17 суток в исследовании динамики с течением времени в клонах (Аг[+]) и (Аг[-]) при высокой репрезентации - по 10000Х клеток на егРНК. Истощение егРНК к целевым генам по сравнению с контрольными егРНК оценивали на протяжении всего исследовании динамики с течением времени как в клонах Аг[+], так и в клонах Аг[-]. Сначала нормализовали число считываний относительно суммы ненацеленных егРНК. Нормализация включала в себя умножение скалярного числа на каждый образец (для разных образцов могли применяться разные скалярные числа) таким образом, что после нормализации общее число считываний по всем ненацеленным егРНК на образец было одинаковым. После такой нормализации ненацеленные егРНК не показали никаких изменений, как и ожидалось, а многие нацеленные егРНК проявили паттерны истощения во времени. При использовании нормализации по сумме ненацеленных егРНК только небольшое число ненацеленных егРНК проявили нарушение (критерии нарушения: значение р≤0,01; кратность изменения ≥1,2). Паттерн был случайным, а величина была небольшой. Помимо визуального (качественного) отображения нарушений в ненацеленных егРНК, с помощью статистического анализа (р-значение и кратность изменения) было продемонстрировано, что на 7-е сутки по сравнению с 3-й сутками, на 10-е сутки по сравнению с 3-й сутками, на 14-е сутки по сравнению с 3-й сутками и на 17-е сутки по сравнению с 3-й сутками было нарушено только небольшое число ненацеленных егРНК. В дополнение к этому, то, какие из егРНК были нарушены в какой момент времени (по сравнению с 3-й сутками), было довольно случайным, потому что тепловая карта не показывала временного паттерна ни для какой из данных нарушенных егРНК (данные не показаны). Затем 10 предполагаемых «основных» генов были валидированы путем оценки истощения егРНК как в образцах Аг[+], так и в образцах Аг[-]. Данные о динамике с течением времени для репликатов егРНК к десяти указанным генам показаны на фиг. 19А-фиг. 19С.
[00277] Затем оценивали паттерны истощения синтетически летальных генов-кандидатов. Для каждой егРНК рассчитывали значимость (р-значение) временного фактора (два эксперимента использовали как повторы для каждого момента времени). К полученным данным (два повторных эксперимента с пятью моментами времени в каждом (с 3-х по 17-е сутки)) применялась стандартная обобщенная модель линейной регрессии (ОМЛР, англ. «GLM»): у ~ бета * t, где у = число считываний и t = время. Решение ОМЛР привело к получению р-значения для параметра бета, которое определяет, насколько значительно бета отличается от 0 (т.е. значимость временного фактора). В дополнение к этому, каждая егРНК была отмечена, если она имела паттерн истощения в каждом эксперименте (10-е сутки <3-й сутки; И 14-е сутки <7-е сутки, И 17-е сутки <10-е сутки). егРНК отбирали, если они имели значимое р-значение с течением времени (≤1е-3) и паттерн истощения в каждом эксперименте. Далее отбирали егРНК, если они имели различимый паттерн истощения между Аг[+] и Аг[-] (дифференциальный тест на вписанных в модель скоростях экспоненциального распада). Чтобы определить, существует ли различимый паттерн истощения, предпринимались следующие шаги: (1) для каждой егРНК X в клетках Аг[+] и Аг[-] усредняли число считываний из двух повторных экспериментов для каждого момента времени; (2) встраивали данные по пяти моментам времени из п. (1) в модель экспоненциального распада: число считываний = ехр (-скорость * t), чтобы оценить скорость, в результате чего получается ожидаемое значение скорости (например, скорость 1) и связанная с ней стандартная ошибка (например, стандартная ошибка 1); и (3) если [скорость 1] / [скорость 2]≥1,5 или ≤1/1,5, и ([скорость 1] - [скорость 2]) / квадратный корень ([стандартная ошибка 1]2+[стандартная ошибка 2]2)≥1,96 или ≤-1,96, то скорость 1 скорость 2 статистически различаются. Пример различимого паттерна истощения (т.е. более резкого или более четкого паттерна истощения в клетках Аг[+]) показан на фиг. 20. Затем отбирали гены на основании числа отобранных егРНК (2 из 4 суммарных егРНК, нацеленных на ген, или 3 из 6 суммарных егРНК, нацеленных на ген).
[00278] Двадцать семь егРНК были идентифицированы как имеющие более резкие паттерны истощения в Аг[+] по сравнению с Аг[-]. См. фиг. 21. Из них четыре целевых гена были отобраны для дальнейшей валидации (целевой ген 1, целевой ген 2, целевой ген 3 и целевой ген 4) на основании числа отобранных егРНК (2 из 4 суммарных егРНК, нацеленных на ген, или 3 из 6 суммарных егРНК, нацеленных на ген). Валидация для целевых генов 1 и 2 показана на фиг. 22А, а валидация для целевых генов 3 и 4 показана на фиг. 22 В. Указанные 10 основных генов были также валидированы способом вторичного скрининга (данные не показаны).
[00279] Для дальнейшей валидации биосенсорные клетки тау-CFP/тау-YFP, экспрессирующие Cas9 и либо содержащие агрегаты (Аг[+]), либо не содержащие агрегаты (Аг[-]), трансдуцировали егРНК, нацеленными на целевой ген 1, целевой ген 2, и целевой ген 4 с использованием подхода лентивирусной доставки для введения нокаутирующих мутаций в каждый целевой ген (фиг. 25). После размножения в условиях отбора в течение 7 суток оценивали и количественно определяли с помощью проточной цитометрии жизнеспособность клеток и гибель клеток (фиг. 26), а также оценивали и подтверждали снижение экспрессии целевых генов (данные не показаны). егРНК, нацеленные на целевой ген 1, целевой ген 2 и целевой ген 4, вызывали избирательную гибель клеток в популяции клеток Аг[+], но не в популяции клеток Аг[-].
Пример 3. Разработка полногеномной скрининговой платформы CRISPR/Cas9 для идентификации генетических модификаторов агрегации тау-белка, влияющих на жизнеспособность клеток, с использованием библиотеки CRISPR/Cas9 для активации транскрипции
[00280] Чтобы идентифицировать гены, которые при активации их транскрипции проявляют синтетическую летальность с ассоциированными с заболеванием белковыми агрегатами, была разработана платформа для проведения полногеномных скринингов с использованием библиотек егРНК активации транскрипции CRISPRa (hSAM). Указанная платформа идентифицирует гены, активация транскрипции которых вызывает гибель клеток, особенно в контексте аномальной агрегации белка. Идентификация таких генов может пролить свет на механизмы нейротоксичности, связанной с агрегатами, и генетические пути, которые способствуют гибели нейронов в контексте нейродегенеративного заболевания.
[00281] В данном скрининге использовалась линия клеток-биосенсоров тау-белка человека, состоящая из клеток НЕК293Т, стабильно экспрессирующих домен тау-белка с четырьмя повторами, tau_4RD, содержащий домен связывания микротрубочек (MBD) тау-белка с патогенной мутацией P301S, слитый либо с CFP, либо с YFP. Это значит, что указанные клеточные линии НЕК293Т содержат два трансгена, стабильно экспрессирующих варианты ассоциированного с заболеванием белка, слитые с флуоресцентным белком CFP или с флуоресцентным белком YFP - тау4RD-CFP/тау4RD-YFP (TCY), где содержащий повторы домен тау-белка (4RD) содержит патогенную мутацию P301S. См. фиг. 1. В данных биосенсорных линиях агрегация белков тау-CFP/тау-YFP производит FRET-сигнал, который является результатом переноса энергии флуоресценции от донора CFP к акцептору YFP. См. фиг. 2. Клетки, положительные в отношении FRET, содержащие агрегаты тау-белка, могут быть отсортированы и выделены с помощью метода проточной цитометрии. В исходном состоянии нестимулированные клетки экспрессируют репортеры в стабильном растворимом виде с минимальным сигналом FRET. При стимуляции (например, липосомная трансфекция засеваемыми частицами) репортерные белки образуют агрегаты, производя сигнал FRET. Клетки, содержащие агрегаты, можно выделить с помощью метода FACS. Стабильно размножающиеся клеточные линии, содержащие агрегаты, Аг[+], могут быть выделены путем клонального серийного разведения клеточных линий Аг[-].
[00282] В данную линию клеток-биосенсоров тау-белка было внесено несколько модификаций, чтобы сделать ее пригодной для генетического скрининга. Сначала данную линию клеток-биосенсоров тау-белка дополнительно трансгенно модифицировали для экспрессии компонентов системы активации транскрипции CRISPR/Cas SAM: dCas9-VP64 и MS2-P65-HSF1. Лентивирусные конструкции dCas9-VP64 и MS2-P65-HSF1 представлены в SEQ ID NO: 42 и 43 соответственно. Клон DC11 был выбран как высокопроизводительный клон для применения в последующих скринингах библиотек. Данный клон был подтвержден на предмет его эффективности в активации выбранных целевых генов.
[00283] Затем были получены субклоны этих SAM-экспрессирующих биосенсорных линий тау-CFP/ray-YFP (TCY), в которых тау-белок стабильно присутствует либо в неагрегированном состоянии (состояние по умолчанию) (Аг[-]), либо в агрегированном состоянии (Аг[+]). Для получения клеточных линий, в которых тау-белок стабильно и постоянно присутствует в агрегированном состоянии, клетки, экспрессирующие SAM, обрабатывали рекомбинантным фибриллизованным тау-белком, смешанным с липофектаминовым реагентом, чтобы «засеять» агрегацию тау-белка, трансгенно экспрессируемого данными клетками. Затем «засеянные» клетки серийно разбавляли для получения клонов, полученных из отдельных клеток, и данные клоны затем размножали для идентификации клональных клеточных линий, в которых агрегаты тау-белка стабильно сохраняются во всех клетках при росте и множественных пассажах с течением времени. Один из данных стабильных клонов Аг[+], положительных в отношении агрегации, DC11-В6 был выбран для размножения и применения в скрининге.
Пример 4. Полногеномная скрининговая платформа CRISPR/Cas9 для идентификации генетических модификаторов агрегации тау-белка, влияющих на жизнеспособность клеток, с использованием библиотеки CRISPR/Cas9 для активации транскрипции
[00284] Для выявления генетических модификаторов тау-белка, влияющих на жизнеспособность клеток, был проведен объединенный полногеномный скрининг CRISPRa активации транскрипции (hSAM) с использованием линий клональных клеток, разработанных в примере 3. Более конкретно, биосенсорные клетки тау-СБР/тау-YFP, экспрессирующие SAM (например, экспрессирующие dCas9-VP64/MS2-P65-HSFl) и либо содержащие агрегаты (Аг[+]), либо не содержащие агрегаты (Аг[-]), трансдуцировали полногеномной библиотекой CRISPR hSAM егРНК человека с использованием подхода лентивирусной доставки для активации транскрипции каждого целевого гена в количестве 500Х клеток на егРНК. Указанная библиотека CRISPR hSAM егРНК нацелена на сайты, расположенные в пределах 200 п. о. в направлении к 5' от сайта начала транскрипции со средним охватом 3-4 егРНК на ген. Указанные егРНК были разработаны таким образом, чтобы избежать нецелевых эффектов, избегая егРНК с двумя или меньшим числом несовпадений с нецелевыми геномными последовательностями. Указанная библиотека охватывает 18946 генов человека. Указанную библиотеку вводили путем трансдукции с показателем множественности заражения (МЗ)<0,3 при охвате >500 клеток на егРНК. Клетки-биосенсоры тау-белка выращивали в условиях отбора зеоцином для отбора клеток с интеграцией и экспрессией уникальной егРНК на клетку.
[00285] Образцы клеток (Аг[+]) и (Аг[-]) собирали в пяти моментах времени: 3-й, 6-е, 7-е, 14-е и 17-е сутки после трансдукции. Выделение ДНК и ПЦР-амплификация интегрированных конструкций егРНК позволили провести характеризацию репертуара егРНК в каждой линии клеток в каждый момент времени с помощью секвенирования нового поколения (СНП). Данный скрининг состоял из 4-х повторных экспериментов. Анализ СНП-данных позволил идентифицировать гены, которые при активации их транскрипции приводят к гибели клеток. егРНК, которые истощены в обеих клеточных линиях, по всей видимости, нарушают некоторые клеточные процессы, необходимые для жизнеспособности клеток, в то время как егРНК, которые специфически истощены в клеточной линии Аг[+], но при этом не истощены или истощены в меньшей степени в клеточной линии Аг[-], могут указывать на синтетический летальный эффект, при котором активация конкретного целевого гена сочетается с наличием агрегатов тау-белка в клетке, чтобы вызвать гибель данной клетки. Эти синтетические летальные гены представляют интерес как потенциальные модификаторы ассоциированной с тау-белком клеточной токсичности.
[00286] Указанный анализ данных отражает подход, использованный в примере 2, и включает в себя: (1) определение профилей егРНК в двух популяциях клеток Аг[+] и Аг[-] в течение 17 суток и (2) сравнение профилей егРНК в Аг[+] по сравнению с Аг[-] в разные моменты времени или в конечный момент времени по сравнению с 3-й сутками. Поскольку указанный анализ данных отражает подход, использованный в примере 2, не все из соответствующих подробностей примера 2 повторяются в данном примере.
[00287] Как объясняется в примере 2, это новый аналитический подход к оценке истощения егРНК по сравнению с более традиционным подходом, заключающимся в простом сравнении СНП-считываний коллекции конечных клеток с первым пассажем. Для сравнения, другие опубликованные полногеномные скрининги CRISPR на предмет истощения сравнивали показатели CRISPR как отношение числа считываний между финальным сбором и ранним пассажем. Напротив, наш подход состоял в том, чтобы изучить полную динамику во времени с целью идентификации генов, которые проявляют паттерн истощения егРНК с течением времени.
[00288] Истощенные гены затем идентифицировали с помощью анализа динамики с течением времени. В каждом повторе отбирали истощенные егРНК с невозрастающими временными паттернами (число считываний на 10-е сутки ≤ на 3-й сутки; число считываний на 14-е сутки ≤ на 7-е сутки; число считываний на 17-е сутки ≤ на 10-е сутки) относительно 3-х суток. Сохраняли егРНК, уже истощенные на 3-й сутки (т.е. те, которые были ниже предела обнаружения, равного 30, на 3-й сутки, и которые оставались ниже предела обнаружения в остальные моменты времени). Флуктуации, близкие к пределу обнаружения, игнорировали (т.е. если число считываний для данной егРНК было меньшим или равным среднему числу считываний егРНК, которые были ниже предела обнаружения + 2 * стандартное отклонение числа считываний егРНК, которые были ниже предела обнаружения, то такую егРНК считали необнаруженной). Гены отбирали, если для них имелось достаточное число истощенных егРНК (1 из 1 суммарной егРНК, 1 из 2 суммарных егРНК, 1 из 3 суммарных егРНК, 2 из 4 суммарных егРНК, 2 из 5 суммарных егРНК или 3 из 6 суммарных егРНК). Затем гены идентифицировали как «основные», если егРНК, нацеленные на данные гены, были истощены как в Аг[+], так и в Аг[-]. Гены идентифицировали как «синтетически летальные», если егРНК, нацеленные на данные гены, были истощены в Аг[+], но не в Аг[-] (отсутствие истощения в Аг[-] по сравнению с Аг[+]; отсеивание на основании нескольких повторов и ручной проверки). Под отсеиванием подразумевается сбор генов, представленных во всех трех повторных экспериментах по исследованию динамики с течением времени в клеточной линии Аг[+], а затем исключение любого гена, представленного по меньшей мере в одном эксперименте в клеточной линии Аг[-]. Ручная проверка включала в себя просмотр гидовых РНК, истощенных в одном из экспериментов с Аг[+], поиск гидовых РНК, истощенных во всех экспериментах с Аг[+], но ни в одном из экспериментов с Аг[-] или только в одном из экспериментов с Аг[-], и подтверждение того, что ни одна цель не была пропущена.
[00289] Гены, на которые нацелены множественные егРНК, проявляющие паттерн истощения при множественных повторных скринингах в Аг[+], но не в Аг[-], были идентифицированы как «синтетически летальные» гены-кандидаты. Затем оценивали паттерны истощения синтетически летальных генов-кандидатов. Для каждой егРНК рассчитывали значимость (р-значение) временного фактора (два эксперимента использовали как повторы для каждого момента времени). К полученным данным (два повторных эксперимента с пятью моментами времени в каждом (с 3-х по 17-е сутки)) применялась стандартная обобщенная модель линейной регрессии (ОМЛР, англ. «GLM»): у ~ бета * t, где у = число считываний и t = время. Решение ОМЛР привело к получению р-значения для параметра бета, которое определяет, насколько значительно бета отличается от 0 (т.е. значимость временного фактора). В дополнение к этому, каждая егРНК была отмечена, если она имела паттерн истощения в каждом эксперименте (10-е сутки <3-й сутки; И 14-е сутки <7-е сутки, И 17-е сутки <10-е сутки). егРНК отбирали, если они имели значимое р-значение с течением времени (≤1е-3) и паттерн истощения в каждом эксперименте. Далее отбирали егРНК, если они имели различимый паттерн истощения между Аг[+] и Аг[-] (дифференциальный тест на вписанных в модель скоростях экспоненциального распада). Чтобы определить, существует ли различимый паттерн истощения, предпринимались следующие шаги: (1) для каждой егРНК X в клетках Аг[+] и Аг[-] усредняли число считываний из двух повторных экспериментов для каждого момента времени; (2) встраивали данные по пяти моментам времени из п. (1) в модель экспоненциального распада: число считываний = ехр (-скорость * t), чтобы оценить скорость, в результате чего получается ожидаемое значение скорости (например, скорость 1) и связанная с ней стандартная ошибка (например, стандартная ошибка 1); и (3) если [скорость 1] / [скорость 2]≥1,5 или ≤1/1,5, и ([скорость 1] - [скорость 2]) / квадратный корень ([стандартная ошибка 1]2+[стандартная ошибка 2]2)≥1,96 или ≤-1,96, то скорость 1 скорость 2 статистически различаются. Пример различимого паттерна истощения (т.е. более резкого или более четкого паттерна истощения в клетках Аг[+]) показан на фиг. 20.
[00290] Тринадцать егРНК, представляющие девять целевых генов (целевые гены 7-15), были идентифицированы как имеющие более резкие паттерны истощения в Аг[+] по сравнению с Аг[-]. См. фиг. 23. Валидация для целевых генов 7-15 показана на фиг. 24А - фиг. 24 В.
--->
SEQUENCE LISTING
<110> Regeneron Pharmaceuticals, Inc.
<120> ПЛАТФОРМА CRISPR/CAS ДЛЯ ИСКЛЮЧАЮЩЕГО СКРИНИНГА ДЛЯ ВЫЯВЛЕНИЯ
ГЕНЕТИЧЕСКИХ УЯЗВИМОСТЕЙ, СВЯЗАННЫХ С АГРЕГАЦИЕЙ ТАУ-БЕЛКА
<130> 057766-544640
<150> US 62/820,101
<151> 2019-03-18
<160> 43
<170> PatentIn, версия 3.5
<210> 1
<211> 31
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 1
Gln Thr Ala Pro Val Pro Met Pro Asp Leu Lys Asn Val Lys Ser Lys
1 5 10 15
Ile Gly Ser Thr Glu Asn Leu Lys His Gln Pro Gly Gly Gly Lys
20 25 30
<210> 2
<211> 31
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 2
Val Gln Ile Ile Asn Lys Lys Leu Asp Leu Ser Asn Val Gln Ser Lys
1 5 10 15
Cys Gly Ser Lys Asp Asn Ile Lys His Val Pro Gly Gly Gly Ser
20 25 30
<210> 3
<211> 31
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 3
Val Gln Ile Val Tyr Lys Pro Val Asp Leu Ser Lys Val Thr Ser Lys
1 5 10 15
Cys Gly Ser Leu Gly Asn Ile His His Lys Pro Gly Gly Gly Gln
20 25 30
<210> 4
<211> 32
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 4
Val Glu Val Lys Ser Glu Lys Leu Asp Phe Lys Asp Arg Val Gln Ser
1 5 10 15
Lys Ile Gly Ser Leu Asp Asn Ile Thr His Val Pro Gly Gly Gly Asn
20 25 30
<210> 5
<211> 93
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 5
cagacagccc ccgtgcccat gccagacctg aagaatgtca agtccaagat cggctccact
60
gagaacctga agcaccagcc gggaggcggg aag
93
<210> 6
<211> 93
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 6
gtgcagataa ttaataagaa gctggatctt agcaacgtcc agtccaagtg tggctcaaag
60
gataatatca aacacgtccc gggaggcggc agt
93
<210> 7
<211> 93
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 7
gtgcaaatag tctacaaacc agttgacctg agcaaggtga cctccaagtg tggctcatta
60
ggcaacatcc atcataaacc aggaggtggc cag
93
<210> 8
<211> 96
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 8
gtggaagtaa aatctgagaa gcttgacttc aaggacagag tccagtcgaa gattgggtcc
60
ctggacaata tcacccacgt ccctggcgga ggaaat
96
<210> 9
<211> 133
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 9
Leu Gln Thr Ala Pro Val Pro Met Pro Asp Leu Lys Asn Val Lys Ser
1 5 10 15
Lys Ile Gly Ser Thr Glu Asn Leu Lys His Gln Pro Gly Gly Gly Lys
20 25 30
Val Gln Ile Ile Asn Lys Lys Leu Asp Leu Ser Asn Val Gln Ser Lys
35 40 45
Cys Gly Ser Lys Asp Asn Ile Lys His Val Pro Gly Gly Gly Ser Val
50 55 60
Gln Ile Val Tyr Lys Pro Val Asp Leu Ser Lys Val Thr Ser Lys Cys
65 70 75 80
Gly Ser Leu Gly Asn Ile His His Lys Pro Gly Gly Gly Gln Val Glu
85 90 95
Val Lys Ser Glu Lys Leu Asp Phe Lys Asp Arg Val Gln Ser Lys Ile
100 105 110
Gly Ser Leu Asp Asn Ile Thr His Val Pro Gly Gly Gly Asn Lys Lys
115 120 125
Ile Glu Thr His Lys
130
<210> 10
<211> 399
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 10
ctgcagacag cccccgtgcc catgccagac ctgaagaatg tcaagtccaa gatcggctcc
60
actgagaacc tgaagcacca gccgggaggc gggaaggtgc agataattaa taagaagctg
120
gatcttagca acgtccagtc caagtgtggc tcaaaggata atatcaaaca cgtcccggga
180
ggcggcagtg tgcaaatagt ctacaaacca gttgacctga gcaaggtgac ctccaagtgt
240
ggctcattag gcaacatcca tcataaacca ggaggtggcc aggtggaagt aaaatctgag
300
aagcttgact tcaaggacag agtccagtcg aagattgggt ccctggacaa tatcacccac
360
gtccctggcg gaggaaataa aaagattgaa acccacaag
399
<210> 11
<211> 133
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 11
Leu Gln Thr Ala Pro Val Pro Met Pro Asp Leu Lys Asn Val Lys Ser
1 5 10 15
Lys Ile Gly Ser Thr Glu Asn Leu Lys His Gln Pro Gly Gly Gly Lys
20 25 30
Val Gln Ile Ile Asn Lys Lys Leu Asp Leu Ser Asn Val Gln Ser Lys
35 40 45
Cys Gly Ser Lys Asp Asn Ile Lys His Val Ser Gly Gly Gly Ser Val
50 55 60
Gln Ile Val Tyr Lys Pro Val Asp Leu Ser Lys Val Thr Ser Lys Cys
65 70 75 80
Gly Ser Leu Gly Asn Ile His His Lys Pro Gly Gly Gly Gln Val Glu
85 90 95
Val Lys Ser Glu Lys Leu Asp Phe Lys Asp Arg Val Gln Ser Lys Ile
100 105 110
Gly Ser Leu Asp Asn Ile Thr His Val Pro Gly Gly Gly Asn Lys Lys
115 120 125
Ile Glu Thr His Lys
130
<210> 12
<211> 399
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 12
ctgcagacag cccccgtgcc catgccagac ctgaagaatg tcaagtccaa gatcggctcc
60
actgagaacc tgaagcacca gccgggaggc gggaaggtgc agataattaa taagaagctg
120
gatcttagca acgtccagtc caagtgtggc tcaaaggata atatcaaaca cgtctcggga
180
ggcggcagtg tgcaaatagt ctacaaacca gttgacctga gcaaggtgac ctccaagtgt
240
ggctcattag gcaacatcca tcataaacca ggaggtggcc aggtggaagt aaaatctgag
300
aagcttgact tcaaggacag agtccagtcg aagattgggt ccctggacaa tatcacccac
360
gtccctggcg gaggaaataa aaagattgaa acccacaag
399
<210> 13
<211> 238
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 13
Val Ser Lys Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu Val
1 5 10 15
Glu Leu Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly Glu
20 25 30
Gly Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile Cys
35 40 45
Thr Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr Leu
50 55 60
Thr Trp Gly Val Gln Cys Phe Ser Arg Tyr Pro Asp His Met Lys Gln
65 70 75 80
His Asp Phe Phe Lys Ser Ala Met Pro Glu Gly Tyr Val Gln Glu Arg
85 90 95
Thr Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys Thr Arg Ala Glu Val
100 105 110
Lys Phe Glu Gly Asp Thr Leu Val Asn Arg Ile Glu Leu Lys Gly Ile
115 120 125
Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly His Lys Leu Glu Tyr Asn
130 135 140
Tyr Ile Ser His Asn Val Tyr Ile Thr Ala Asp Lys Gln Lys Asn Gly
145 150 155 160
Ile Lys Ala Asn Phe Lys Ile Arg His Asn Ile Glu Asp Gly Ser Val
165 170 175
Gln Leu Ala Asp His Tyr Gln Gln Asn Thr Pro Ile Gly Asp Gly Pro
180 185 190
Val Leu Leu Pro Asp Asn His Tyr Leu Ser Thr Gln Ser Ala Leu Ser
195 200 205
Lys Asp Pro Asn Glu Lys Arg Asp His Met Val Leu Leu Glu Phe Val
210 215 220
Thr Ala Ala Gly Ile Thr Leu Gly Met Asp Glu Leu Tyr Lys
225 230 235
<210> 14
<211> 714
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 14
gtgagcaagg gcgaggagct gttcaccggg gtggtgccca tcctggtcga gctggacggc
60
gacgtaaacg gccacaagtt cagcgtgtcc ggcgagggcg agggcgatgc cacctacggc
120
aagctgaccc tgaagttcat ctgcaccacc ggcaagctgc ccgtgccctg gcccaccctc
180
gtgaccaccc tgacctgggg cgtgcagtgc ttcagccgct accccgacca catgaagcag
240
cacgacttct tcaagtccgc catgcccgaa ggctacgtcc aggagcgcac catcttcttc
300
aaggacgacg gcaactacaa gacccgcgcc gaggtgaagt tcgagggcga caccctggtg
360
aaccgcatcg agctgaaggg catcgacttc aaggaggacg gcaacatcct ggggcacaag
420
ctggagtaca actacatcag ccacaacgtc tatatcaccg ccgacaagca gaagaacggc
480
atcaaggcca acttcaagat ccgccacaac atcgaggacg gcagcgtgca gctcgccgac
540
cactaccagc agaacacccc catcggcgac ggccccgtgc tgctgcccga caaccactac
600
ctgagcaccc agtccgccct gagcaaagac cccaacgaga agcgcgatca catggtcctg
660
ctggagttcg tgaccgccgc cgggatcact ctcggcatgg acgagctgta caag
714
<210> 15
<211> 236
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 15
Val Ser Lys Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu Val
1 5 10 15
Glu Leu Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly Glu
20 25 30
Gly Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile Cys
35 40 45
Thr Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr Phe
50 55 60
Gly Tyr Gly Leu Gln Cys Phe Ala Arg Tyr Pro Asp His Met Lys Gln
65 70 75 80
His Asp Phe Phe Lys Ser Ala Met Pro Glu Gly Tyr Val Gln Glu Arg
85 90 95
Thr Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys Thr Arg Ala Glu Val
100 105 110
Lys Phe Glu Gly Asp Thr Leu Val Asn Arg Ile Glu Leu Lys Gly Ile
115 120 125
Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly His Lys Leu Glu Tyr Asn
130 135 140
Tyr Asn Ser His Asn Val Tyr Ile Met Ala Asp Lys Gln Lys Asn Gly
145 150 155 160
Ile Lys Val Asn Phe Lys Ile Arg His Asn Ile Glu Asp Gly Ser Val
165 170 175
Gln Leu Ala Asp His Tyr Gln Gln Asn Thr Pro Ile Gly Asp Gly Pro
180 185 190
Val Leu Leu Pro Asp Asn His Tyr Leu Ser Tyr Gln Ser Ala Leu Ser
195 200 205
Lys Asp Pro Asn Glu Lys Arg Asp His Met Val Leu Leu Glu Phe Val
210 215 220
Thr Ala Ala Gly Ile Thr Leu Gly Met Asp Glu Leu
225 230 235
<210> 16
<211> 708
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 16
gtgagcaagg gcgaggagct gttcaccggg gtggtgccca tcctggtcga gctggacggc
60
gacgtaaacg gccacaagtt cagcgtgtcc ggcgagggcg agggcgatgc cacctacggc
120
aagctgaccc tgaagttcat ctgcaccacc ggcaagctgc ccgtgccctg gcccaccctc
180
gtgaccacct tcggctacgg cctgcagtgc ttcgcccgct accccgacca catgaagcag
240
cacgacttct tcaagtccgc catgcccgaa ggctacgtcc aggagcgcac catcttcttc
300
aaggacgacg gcaactacaa gacccgcgcc gaggtgaagt tcgagggcga caccctggtg
360
aaccgcatcg agctgaaggg catcgacttc aaggaggacg gcaacatcct ggggcacaag
420
ctggagtaca actacaacag ccacaacgtc tatatcatgg ccgacaagca gaagaacggc
480
atcaaggtga acttcaagat ccgccacaac atcgaggacg gcagcgtgca gctcgccgac
540
cactaccagc agaacacccc catcggcgac ggccccgtgc tgctgcccga caaccactac
600
ctgagctacc agtccgccct gagcaaagac cccaacgaga agcgcgatca catggtcctg
660
ctggagttcg tgaccgccgc cgggatcact ctcggcatgg acgagctg
708
<210> 17
<211> 77
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 17
guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc cguuaucaac uugaaaaagu
60
ggcaccgagu cggugcu
77
<210> 18
<211> 82
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 18
guuggaacca uucaaaacag cauagcaagu uaaaauaagg cuaguccguu aucaacuuga
60
aaaaguggca ccgagucggu gc
82
<210> 19
<211> 76
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 19
guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc cguuaucaac uugaaaaagu
60
ggcaccgagu cggugc
76
<210> 20
<211> 86
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 20
guuuaagagc uaugcuggaa acagcauagc aaguuuaaau aaggcuaguc cguuaucaac
60
uugaaaaagu ggcaccgagu cggugc
86
<210> 21
<211> 1392
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<220>
<221> ПРОЧИЙ_ПРИЗНАК
<222> (1)..(1384)
<223> Cas9
<220>
<221> ПРОЧИЙ_ПРИЗНАК
<222> (1385)..(1392)
<223> FLAG
<400> 21
Met Asp Lys Lys Tyr Ser Ile Gly Leu Asp Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala
1010 1015 1020
Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe
1025 1030 1035
Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala
1040 1045 1050
Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu
1055 1060 1065
Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val
1070 1075 1080
Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys Thr
1085 1090 1095
Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro Lys
1100 1105 1110
Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp Pro
1115 1120 1125
Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser Val
1130 1135 1140
Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu Lys
1145 1150 1155
Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser Ser
1160 1165 1170
Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr Lys
1175 1180 1185
Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser Leu
1190 1195 1200
Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala Gly
1205 1210 1215
Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr Val
1220 1225 1230
Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys
1250 1255 1260
His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys
1265 1270 1275
Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala
1280 1285 1290
Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn
1295 1300 1305
Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala
1310 1315 1320
Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser
1325 1330 1335
Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile Thr
1340 1345 1350
Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly Asp
1355 1360 1365
Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys Lys
1370 1375 1380
Lys Asp Tyr Lys Asp Asp Asp Asp Lys
1385 1390
<210> 22
<211> 4176
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<220>
<221> прочий_признак
<222> (1)..(4152)
<223> Cas9
<220>
<221> прочий_признак
<222> (4153)..(4176)
<223> FLAG
<400> 22
atggacaaga agtacagcat cggcctggac atcggcacca actctgtggg ctgggccgtg
60
atcaccgacg agtacaaggt gcccagcaag aaattcaagg tgctgggcaa caccgaccgg
120
cacagcatca agaagaacct gatcggagcc ctgctgttcg acagcggcga aacagccgag
180
gccacccggc tgaagagaac cgccagaaga agatacacca gacggaagaa ccggatctgc
240
tatctgcaag agatcttcag caacgagatg gccaaggtgg acgacagctt cttccacaga
300
ctggaagagt ccttcctggt ggaagaggat aagaagcacg agcggcaccc catcttcggc
360
aacatcgtgg acgaggtggc ctaccacgag aagtacccca ccatctacca cctgagaaag
420
aaactggtgg acagcaccga caaggccgac ctgcggctga tctatctggc cctggcccac
480
atgatcaagt tccggggcca cttcctgatc gagggcgacc tgaaccccga caacagcgac
540
gtggacaagc tgttcatcca gctggtgcag acctacaacc agctgttcga ggaaaacccc
600
atcaacgcca gcggcgtgga cgccaaggcc atcctgtctg ccagactgag caagagcaga
660
cggctggaaa atctgatcgc ccagctgccc ggcgagaaga agaatggcct gttcggaaac
720
ctgattgccc tgagcctggg cctgaccccc aacttcaaga gcaacttcga cctggccgag
780
gatgccaaac tgcagctgag caaggacacc tacgacgacg acctggacaa cctgctggcc
840
cagatcggcg accagtacgc cgacctgttt ctggccgcca agaacctgtc cgacgccatc
900
ctgctgagcg acatcctgag agtgaacacc gagatcacca aggcccccct gagcgcctct
960
atgatcaaga gatacgacga gcaccaccag gacctgaccc tgctgaaagc tctcgtgcgg
1020
cagcagctgc ctgagaagta caaagagatt ttcttcgacc agagcaagaa cggctacgcc
1080
ggctacattg acggcggagc cagccaggaa gagttctaca agttcatcaa gcccatcctg
1140
gaaaagatgg acggcaccga ggaactgctc gtgaagctga acagagagga cctgctgcgg
1200
aagcagcgga ccttcgacaa cggcagcatc ccccaccaga tccacctggg agagctgcac
1260
gccattctgc ggcggcagga agatttttac ccattcctga aggacaaccg ggaaaagatc
1320
gagaagatcc tgaccttccg catcccctac tacgtgggcc ctctggccag gggaaacagc
1380
agattcgcct ggatgaccag aaagagcgag gaaaccatca ccccctggaa cttcgaggaa
1440
gtggtggaca agggcgcttc cgcccagagc ttcatcgagc ggatgaccaa cttcgataag
1500
aacctgccca acgagaaggt gctgcccaag cacagcctgc tgtacgagta cttcaccgtg
1560
tataacgagc tgaccaaagt gaaatacgtg accgagggaa tgagaaagcc cgccttcctg
1620
agcggcgagc agaaaaaggc catcgtggac ctgctgttca agaccaaccg gaaagtgacc
1680
gtgaagcagc tgaaagagga ctacttcaag aaaatcgagt gcttcgactc cgtggaaatc
1740
tccggcgtgg aagatcggtt caacgcctcc ctgggcacat accacgatct gctgaaaatt
1800
atcaaggaca aggacttcct ggacaatgag gaaaacgagg acattctgga agatatcgtg
1860
ctgaccctga cactgtttga ggacagagag atgatcgagg aacggctgaa aacctatgcc
1920
cacctgttcg acgacaaagt gatgaagcag ctgaagcggc ggagatacac cggctggggc
1980
aggctgagcc ggaagctgat caacggcatc cgggacaagc agtccggcaa gacaatcctg
2040
gatttcctga agtccgacgg cttcgccaac agaaacttca tgcagctgat ccacgacgac
2100
agcctgacct ttaaagagga catccagaaa gcccaggtgt ccggccaggg cgatagcctg
2160
cacgagcaca ttgccaatct ggccggcagc cccgccatta agaagggcat cctgcagaca
2220
gtgaaggtgg tggacgagct cgtgaaagtg atgggccggc acaagcccga gaacatcgtg
2280
atcgaaatgg ccagagagaa ccagaccacc cagaagggac agaagaacag ccgcgagaga
2340
atgaagcgga tcgaagaggg catcaaagag ctgggcagcc agatcctgaa agaacacccc
2400
gtggaaaaca cccagctgca gaacgagaag ctgtacctgt actacctgca gaatgggcgg
2460
gatatgtacg tggaccagga actggacatc aaccggctgt ccgactacga tgtggaccat
2520
atcgtgcctc agagctttct gaaggacgac tccatcgaca acaaggtgct gaccagaagc
2580
gacaagaacc ggggcaagag cgacaacgtg ccctccgaag aggtcgtgaa gaagatgaag
2640
aactactggc ggcagctgct gaacgccaag ctgattaccc agagaaagtt cgacaatctg
2700
accaaggccg agagaggcgg cctgagcgaa ctggataagg ccggcttcat caagagacag
2760
ctggtggaaa cccggcagat cacaaagcac gtggcacaga tcctggactc ccggatgaac
2820
actaagtacg acgagaatga caagctgatc cgggaagtga aagtgatcac cctgaagtcc
2880
aagctggtgt ccgatttccg gaaggatttc cagttttaca aagtgcgcga gatcaacaac
2940
taccaccacg cccacgacgc ctacctgaac gccgtcgtgg gaaccgccct gatcaaaaag
3000
taccctaagc tggaaagcga gttcgtgtac ggcgactaca aggtgtacga cgtgcggaag
3060
atgatcgcca agagcgagca ggaaatcggc aaggctaccg ccaagtactt cttctacagc
3120
aacatcatga actttttcaa gaccgagatt accctggcca acggcgagat ccggaagcgg
3180
cctctgatcg agacaaacgg cgaaaccggg gagatcgtgt gggataaggg ccgggatttt
3240
gccaccgtgc ggaaagtgct gagcatgccc caagtgaata tcgtgaaaaa gaccgaggtg
3300
cagacaggcg gcttcagcaa agagtctatc ctgcccaaga ggaacagcga taagctgatc
3360
gccagaaaga aggactggga ccctaagaag tacggcggct tcgacagccc caccgtggcc
3420
tattctgtgc tggtggtggc caaagtggaa aagggcaagt ccaagaaact gaagagtgtg
3480
aaagagctac tggggatcac catcatggaa agaagcagct tcgagaagaa tcccatcgac
3540
tttctggaag ccaagggcta caaagaagtg aaaaaggacc tgatcatcaa gctgcctaag
3600
tactccctgt tcgagctgga aaacggccgg aagagaatgc tggcctctgc cggcgaactg
3660
cagaagggaa acgaactggc cctgccctcc aaatatgtga acttcctgta cctggccagc
3720
cactatgaga agctgaaggg ctcccccgag gataatgagc agaaacagct gtttgtggaa
3780
cagcacaagc actacctgga cgagatcatc gagcagatca gcgagttctc caagagagtg
3840
atcctggccg acgctaatct ggacaaagtg ctgtccgcct acaacaagca ccgggataag
3900
cccatcagag agcaggccga gaatatcatc cacctgttta ccctgaccaa tctgggagcc
3960
cctgccgcct tcaagtactt tgacaccacc atcgaccgga agaggtacac cagcaccaaa
4020
gaggtgctgg acgccaccct gatccaccag agcatcaccg gcctgtacga gacacggatc
4080
gacctgtctc agctgggagg cgacaagcga cctgccgcca caaagaaggc tggacaggct
4140
aagaagaaga aagattacaa agacgatgac gataag
4176
<210> 23
<211> 16
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 23
guuuuagagc uaugcu
16
<210> 24
<211> 67
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 24
agcauagcaa guuaaaauaa ggcuaguccg uuaucaacuu gaaaaagugg caccgagucg
60
gugcuuu
67
<210> 25
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<220>
<221> прочий_признак
<222> (2)..(21)
<223> n = A, T, C или G
<400> 25
gnnnnnnnnn nnnnnnnnnn ngg
23
<210> 26
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<220>
<221> прочий_признак
<222> (1)..(21)
<223> n = A, T, C или G
<400> 26
nnnnnnnnnn nnnnnnnnnn ngg
23
<210> 27
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<220>
<221> прочий_признак
<222> (3)..(23)
<223> n = A, T, C или G
<400> 27
ggnnnnnnnn nnnnnnnnnn nnngg
25
<210> 28
<211> 72
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 28
aaacagcaua gcaaguuaaa auaaggcuag uccguuauca acuugaaaaa guggcaccga
60
gucggugcuu uu
72
<210> 29
<211> 82
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 29
guuggaacca uucaaaacag cauagcaagu uaaaauaagg cuaguccguu aucaacuuga
60
aaaaguggca ccgagucggu gc
82
<210> 30
<211> 83
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 30
guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc cguuaucaac uugaaaaagu
60
ggcaccgagu cggugcuuuu uuu
83
<210> 31
<211> 80
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 31
guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc cguuaucaac uugaaaaagu
60
ggcaccgagu cggugcuuuu
80
<210> 32
<211> 92
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 32
guuuaagagc uaugcuggaa acagcauagc aaguuuaaau aaggcuaguc cguuaucaac
60
uugaaaaagu ggcaccgagu cggugcuuuu uu
92
<210> 33
<211> 34
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 33
ggccaacaug aggaucaccc augucugcag ggcc
34
<210> 34
<211> 137
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 34
guuuuagagc uaggccaaca ugaggaucac ccaugucugc agggccuagc aaguuaaaau
60
aaggcuaguc cguuaucaac uuggccaaca ugaggaucac ccaugucugc agggccaagu
120
ggcaccgagu cggugcu
137
<210> 35
<211> 157
<212> РНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<220>
<221> прочий_признак
<222> (1)..(20)
<223> n представляет собой a, c, g или u
<400> 35
nnnnnnnnnn nnnnnnnnnn guuuuagagc uaggccaaca ugaggaucac ccaugucugc
60
agggccuagc aaguuaaaau aaggcuaguc cguuaucaac uuggccaaca ugaggaucac
120
ccaugucugc agggccaagu ggcaccgagu cggugcu
157
<210> 36
<211> 1471
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<220>
<221> ПРОЧИЙ_ПРИЗНАК
<222> (1)..(1384)
<223> dCas9
<220>
<221> ПРОЧИЙ_ПРИЗНАК
<222> (1405)..(1409)
<223> NLS
<220>
<221> ПРОЧИЙ_ПРИЗНАК
<222> (1410)..(1471)
<223> VP64
<400> 36
Met Lys Arg Pro Ala Ala Thr Lys Lys Ala Gly Gln Ala Lys Lys Lys
1 5 10 15
Lys Asp Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val
20 25 30
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
35 40 45
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
50 55 60
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
65 70 75 80
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
85 90 95
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
100 105 110
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
115 120 125
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
130 135 140
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
145 150 155 160
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
165 170 175
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
180 185 190
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
195 200 205
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
210 215 220
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
225 230 235 240
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
245 250 255
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
260 265 270
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
275 280 285
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
290 295 300
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
305 310 315 320
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
325 330 335
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
340 345 350
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
355 360 365
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
370 375 380
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
385 390 395 400
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
405 410 415
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
420 425 430
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
435 440 445
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
450 455 460
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
465 470 475 480
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
485 490 495
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
500 505 510
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
515 520 525
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
530 535 540
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
545 550 555 560
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
565 570 575
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
580 585 590
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
595 600 605
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
610 615 620
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
625 630 635 640
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
645 650 655
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
660 665 670
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
675 680 685
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
690 695 700
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
705 710 715 720
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
725 730 735
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
740 745 750
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
755 760 765
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
770 775 780
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
785 790 795 800
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
805 810 815
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
820 825 830
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
835 840 845
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
850 855 860
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Ala Arg
865 870 875 880
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
885 890 895
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
900 905 910
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
915 920 925
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
930 935 940
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
945 950 955 960
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
965 970 975
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
980 985 990
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
995 1000 1005
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu
1010 1015 1020
Phe Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile
1025 1030 1035
Ala Lys Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe
1040 1045 1050
Phe Tyr Ser Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu
1055 1060 1065
Ala Asn Gly Glu Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly
1070 1075 1080
Glu Thr Gly Glu Ile Val Trp Asp Lys Gly Arg Asp Phe Ala Thr
1085 1090 1095
Val Arg Lys Val Leu Ser Met Pro Gln Val Asn Ile Val Lys Lys
1100 1105 1110
Thr Glu Val Gln Thr Gly Gly Phe Ser Lys Glu Ser Ile Leu Pro
1115 1120 1125
Lys Arg Asn Ser Asp Lys Leu Ile Ala Arg Lys Lys Asp Trp Asp
1130 1135 1140
Pro Lys Lys Tyr Gly Gly Phe Asp Ser Pro Thr Val Ala Tyr Ser
1145 1150 1155
Val Leu Val Val Ala Lys Val Glu Lys Gly Lys Ser Lys Lys Leu
1160 1165 1170
Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile Met Glu Arg Ser
1175 1180 1185
Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala Lys Gly Tyr
1190 1195 1200
Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys Tyr Ser
1205 1210 1215
Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser Ala
1220 1225 1230
Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr
1235 1240 1245
Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly
1250 1255 1260
Ser Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His
1265 1270 1275
Lys His Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser
1280 1285 1290
Lys Arg Val Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser
1295 1300 1305
Ala Tyr Asn Lys His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu
1310 1315 1320
Asn Ile Ile His Leu Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala
1325 1330 1335
Ala Phe Lys Tyr Phe Asp Thr Thr Ile Asp Arg Lys Arg Tyr Thr
1340 1345 1350
Ser Thr Lys Glu Val Leu Asp Ala Thr Leu Ile His Gln Ser Ile
1355 1360 1365
Thr Gly Leu Tyr Glu Thr Arg Ile Asp Leu Ser Gln Leu Gly Gly
1370 1375 1380
Asp Ser Ala Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
1385 1390 1395
Gly Gly Ser Gly Pro Lys Lys Lys Arg Lys Val Ala Ala Ala Gly
1400 1405 1410
Ser Gly Arg Ala Asp Ala Leu Asp Asp Phe Asp Leu Asp Met Leu
1415 1420 1425
Gly Ser Asp Ala Leu Asp Asp Phe Asp Leu Asp Met Leu Gly Ser
1430 1435 1440
Asp Ala Leu Asp Asp Phe Asp Leu Asp Met Leu Gly Ser Asp Ala
1445 1450 1455
Leu Asp Asp Phe Asp Leu Asp Met Leu Ile Asn Cys Thr
1460 1465 1470
<210> 37
<211> 473
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<220>
<221> ПРОЧИЙ_ПРИЗНАК
<222> (1)..(130)
<223> MCP
<220>
<221> ПРОЧИЙ_ПРИЗНАК
<222> (151)..(155)
<223> NLS
<220>
<221> ПРОЧИЙ_ПРИЗНАК
<222> (161)..(341)
<223> P65
<220>
<221> ПРОЧИЙ_ПРИЗНАК
<222> (350)..(473)
<223> HSF1
<400> 37
Met Ala Ser Asn Phe Thr Gln Phe Val Leu Val Asp Asn Gly Gly Thr
1 5 10 15
Gly Asp Val Thr Val Ala Pro Ser Asn Phe Ala Asn Gly Val Ala Glu
20 25 30
Trp Ile Ser Ser Asn Ser Arg Ser Gln Ala Tyr Lys Val Thr Cys Ser
35 40 45
Val Arg Gln Ser Ser Ala Gln Lys Arg Lys Tyr Thr Ile Lys Val Glu
50 55 60
Val Pro Lys Val Ala Thr Gln Thr Val Gly Gly Val Glu Leu Pro Val
65 70 75 80
Ala Ala Trp Arg Ser Tyr Leu Asn Met Glu Leu Thr Ile Pro Ile Phe
85 90 95
Ala Thr Asn Ser Asp Cys Glu Leu Ile Val Lys Ala Met Gln Gly Leu
100 105 110
Leu Lys Asp Gly Asn Pro Ile Pro Ser Ala Ile Ala Ala Asn Ser Gly
115 120 125
Ile Tyr Ser Ala Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
130 135 140
Gly Gly Ser Gly Pro Lys Lys Lys Arg Lys Val Ala Ala Ala Gly Ser
145 150 155 160
Pro Ser Gly Gln Ile Ser Asn Gln Ala Leu Ala Leu Ala Pro Ser Ser
165 170 175
Ala Pro Val Leu Ala Gln Thr Met Val Pro Ser Ser Ala Met Val Pro
180 185 190
Leu Ala Gln Pro Pro Ala Pro Ala Pro Val Leu Thr Pro Gly Pro Pro
195 200 205
Gln Ser Leu Ser Ala Pro Val Pro Lys Ser Thr Gln Ala Gly Glu Gly
210 215 220
Thr Leu Ser Glu Ala Leu Leu His Leu Gln Phe Asp Ala Asp Glu Asp
225 230 235 240
Leu Gly Ala Leu Leu Gly Asn Ser Thr Asp Pro Gly Val Phe Thr Asp
245 250 255
Leu Ala Ser Val Asp Asn Ser Glu Phe Gln Gln Leu Leu Asn Gln Gly
260 265 270
Val Ser Met Ser His Ser Thr Ala Glu Pro Met Leu Met Glu Tyr Pro
275 280 285
Glu Ala Ile Thr Arg Leu Val Thr Gly Ser Gln Arg Pro Pro Asp Pro
290 295 300
Ala Pro Thr Pro Leu Gly Thr Ser Gly Leu Pro Asn Gly Leu Ser Gly
305 310 315 320
Asp Glu Asp Phe Ser Ser Ile Ala Asp Met Asp Phe Ser Ala Leu Leu
325 330 335
Ser Gln Ile Ser Ser Ser Gly Gln Gly Gly Gly Gly Ser Gly Phe Ser
340 345 350
Val Asp Thr Ser Ala Leu Leu Asp Leu Phe Ser Pro Ser Val Thr Val
355 360 365
Pro Asp Met Ser Leu Pro Asp Leu Asp Ser Ser Leu Ala Ser Ile Gln
370 375 380
Glu Leu Leu Ser Pro Gln Glu Pro Pro Arg Pro Pro Glu Ala Glu Asn
385 390 395 400
Ser Ser Pro Asp Ser Gly Lys Gln Leu Val His Tyr Thr Ala Gln Pro
405 410 415
Leu Phe Leu Leu Asp Pro Gly Ser Val Asp Thr Gly Ser Asn Asp Leu
420 425 430
Pro Val Leu Phe Glu Leu Gly Glu Gly Ser Tyr Phe Ser Glu Gly Asp
435 440 445
Gly Phe Ala Glu Asp Pro Thr Ile Ser Leu Leu Thr Gly Ser Glu Pro
450 455 460
Pro Lys Ala Lys Asp Pro Thr Val Ser
465 470
<210> 38
<211> 4413
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<220>
<221> прочий_признак
<222> (1)..(4152)
<223> dCas9
<220>
<221> прочий_признак
<222> (4213)..(4227)
<223> NLS
<220>
<221> прочий_признак
<222> (4228)..(4413)
<223> VP64
<400> 38
atgaaaaggc cggcggccac gaaaaaggcc ggccaggcaa aaaagaaaaa ggacaagaag
60
tacagcatcg gcctggccat cggcaccaac tctgtgggct gggccgtgat caccgacgag
120
tacaaggtgc ccagcaagaa attcaaggtg ctgggcaaca ccgaccggca cagcatcaag
180
aagaacctga tcggagccct gctgttcgac agcggcgaaa cagccgaggc cacccggctg
240
aagagaaccg ccagaagaag atacaccaga cggaagaacc ggatctgcta tctgcaagag
300
atcttcagca acgagatggc caaggtggac gacagcttct tccacagact ggaagagtcc
360
ttcctggtgg aagaggataa gaagcacgag cggcacccca tcttcggcaa catcgtggac
420
gaggtggcct accacgagaa gtaccccacc atctaccacc tgagaaagaa actggtggac
480
agcaccgaca aggccgacct gcggctgatc tatctggccc tggcccacat gatcaagttc
540
cggggccact tcctgatcga gggcgacctg aaccccgaca acagcgacgt ggacaagctg
600
ttcatccagc tggtgcagac ctacaaccag ctgttcgagg aaaaccccat caacgccagc
660
ggcgtggacg ccaaggccat cctgtctgcc agactgagca agagcagacg gctggaaaat
720
ctgatcgccc agctgcccgg cgagaagaag aatggcctgt tcggcaacct gattgccctg
780
agcctgggcc tgacccccaa cttcaagagc aacttcgacc tggccgagga tgccaaactg
840
cagctgagca aggacaccta cgacgacgac ctggacaacc tgctggccca gatcggcgac
900
cagtacgccg acctgtttct ggccgccaag aacctgtccg acgccatcct gctgagcgac
960
atcctgagag tgaacaccga gatcaccaag gcccccctga gcgcctctat gatcaagaga
1020
tacgacgagc accaccagga cctgaccctg ctgaaagctc tcgtgcggca gcagctgcct
1080
gagaagtaca aagagatttt cttcgaccag agcaagaacg gctacgccgg ctacattgac
1140
ggcggagcca gccaggaaga gttctacaag ttcatcaagc ccatcctgga aaagatggac
1200
ggcaccgagg aactgctcgt gaagctgaac agagaggacc tgctgcggaa gcagcggacc
1260
ttcgacaacg gcagcatccc ccaccagatc cacctgggag agctgcacgc cattctgcgg
1320
cggcaggaag atttttaccc attcctgaag gacaaccggg aaaagatcga gaagatcctg
1380
accttccgca tcccctacta cgtgggccct ctggccaggg gaaacagcag attcgcctgg
1440
atgaccagaa agagcgagga aaccatcacc ccctggaact tcgaggaagt ggtggacaag
1500
ggcgcttccg cccagagctt catcgagcgg atgaccaact tcgataagaa cctgcccaac
1560
gagaaggtgc tgcccaagca cagcctgctg tacgagtact tcaccgtgta taacgagctg
1620
accaaagtga aatacgtgac cgagggaatg agaaagcccg ccttcctgag cggcgagcag
1680
aaaaaggcca tcgtggacct gctgttcaag accaaccgga aagtgaccgt gaagcagctg
1740
aaagaggact acttcaagaa aatcgagtgc ttcgactccg tggaaatctc cggcgtggaa
1800
gatcggttca acgcctccct gggcacatac cacgatctgc tgaaaattat caaggacaag
1860
gacttcctgg acaatgagga aaacgaggac attctggaag atatcgtgct gaccctgaca
1920
ctgtttgagg acagagagat gatcgaggaa cggctgaaaa cctatgccca cctgttcgac
1980
gacaaagtga tgaagcagct gaagcggcgg agatacaccg gctggggcag gctgagccgg
2040
aagctgatca acggcatccg ggacaagcag tccggcaaga caatcctgga tttcctgaag
2100
tccgacggct tcgccaacag aaacttcatg cagctgatcc acgacgacag cctgaccttt
2160
aaagaggaca tccagaaagc ccaggtgtcc ggccagggcg atagcctgca cgagcacatt
2220
gccaatctgg ccggcagccc cgccattaag aagggcatcc tgcagacagt gaaggtggtg
2280
gacgagctcg tgaaagtgat gggccggcac aagcccgaga acatcgtgat cgaaatggcc
2340
agagagaacc agaccaccca gaagggacag aagaacagcc gcgagagaat gaagcggatc
2400
gaagagggca tcaaagagct gggcagccag atcctgaaag aacaccccgt ggaaaacacc
2460
cagctgcaga acgagaagct gtacctgtac tacctgcaga atgggcggga tatgtacgtg
2520
gaccaggaac tggacatcaa ccggctgtcc gactacgatg tggaccacat cgtgcctcag
2580
agctttctga aggacgactc catcgacaac aaggtgctga ccagaagcga caaggcccgg
2640
ggcaagagcg acaacgtgcc ctccgaagag gtcgtgaaga agatgaagaa ctactggcgg
2700
cagctgctga acgccaagct gattacccag agaaagttcg acaatctgac caaggccgag
2760
agaggcggcc tgagcgaact ggataaggcc ggcttcatca agagacagct ggtggaaacc
2820
cggcagatca caaagcacgt ggcacagatc ctggactccc ggatgaacac taagtacgac
2880
gagaatgaca agctgatccg ggaagtgaaa gtgatcaccc tgaagtccaa gctggtgtcc
2940
gatttccgga aggatttcca gttttacaaa gtgcgcgaga tcaacaacta ccaccacgcc
3000
cacgacgcct acctgaacgc cgtcgtggga accgccctga tcaaaaagta ccctaagctg
3060
gaaagcgagt tcgtgtacgg cgactacaag gtgtacgacg tgcggaagat gatcgccaag
3120
agcgagcagg aaatcggcaa ggctaccgcc aagtacttct tctacagcaa catcatgaac
3180
tttttcaaga ccgagattac cctggccaac ggcgagatcc ggaagcggcc tctgatcgag
3240
acaaacggcg aaaccgggga gatcgtgtgg gataagggcc gggattttgc caccgtgcgg
3300
aaagtgctga gcatgcccca agtgaatatc gtgaaaaaga ccgaggtgca gacaggcggc
3360
ttcagcaaag agtctatcct gcccaagagg aacagcgata agctgatcgc cagaaagaag
3420
gactgggacc ctaagaagta cggcggcttc gacagcccca ccgtggccta ttctgtgctg
3480
gtggtggcca aagtggaaaa gggcaagtcc aagaaactga agagtgtgaa agagctgctg
3540
gggatcacca tcatggaaag aagcagcttc gagaagaatc ccatcgactt tctggaagcc
3600
aagggctaca aagaagtgaa aaaggacctg atcatcaagc tgcctaagta ctccctgttc
3660
gagctggaaa acggccggaa gagaatgctg gcctctgccg gcgaactgca gaagggaaac
3720
gaactggccc tgccctccaa atatgtgaac ttcctgtacc tggccagcca ctatgagaag
3780
ctgaagggct cccccgagga taatgagcag aaacagctgt ttgtggaaca gcacaagcac
3840
tacctggacg agatcatcga gcagatcagc gagttctcca agagagtgat cctggccgac
3900
gctaatctgg acaaagtgct gtccgcctac aacaagcacc gggataagcc catcagagag
3960
caggccgaga atatcatcca cctgtttacc ctgaccaatc tgggagcccc tgccgccttc
4020
aagtactttg acaccaccat cgaccggaag aggtacacca gcaccaaaga ggtgctggac
4080
gccaccctga tccaccagag catcaccggc ctgtacgaga cacggatcga cctgtctcag
4140
ctgggaggcg acagcgctgg aggaggtgga agcggaggag gaggaagcgg aggaggaggt
4200
agcggaccta agaaaaagag gaaggtggcg gccgctggat ccggacgggc tgacgcattg
4260
gacgattttg atctggatat gctgggaagt gacgccctcg atgattttga ccttgacatg
4320
cttggttcgg atgcccttga tgactttgac ctcgacatgc tcggcagtga cgcccttgat
4380
gatttcgacc tggacatgct gattaactgt aca
4413
<210> 39
<211> 1419
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<220>
<221> прочий_признак
<222> (1)..(390)
<223> MCP
<220>
<221> прочий_признак
<222> (451)..(465)
<223> NLS
<220>
<221> прочий_признак
<222> (481)..(1023)
<223> P65
<220>
<221> прочий_признак
<222> (1048)..(1419)
<223> HSF1
<400> 39
atggcttcaa actttactca gttcgtgctc gtggacaatg gtgggacagg ggatgtgaca
60
gtggctcctt ctaatttcgc taatggggtg gcagagtgga tcagctccaa ctcacggagc
120
caggcctaca aggtgacatg cagcgtcagg cagtctagtg cccagaagag aaagtatacc
180
atcaaggtgg aggtccccaa agtggctacc cagacagtgg gcggagtcga actgcctgtc
240
gccgcttgga ggtcctacct gaacatggag ctcactatcc caattttcgc taccaattct
300
gactgtgaac tcatcgtgaa ggcaatgcag gggctcctca aagacggtaa tcctatccct
360
tccgccatcg ccgctaactc aggtatctac agcgctggag gaggtggaag cggaggagga
420
ggaagcggag gaggaggtag cggacctaag aaaaagagga aggtggcggc cgctggatcc
480
ccttcagggc agatcagcaa ccaggccctg gctctggccc ctagctccgc tccagtgctg
540
gcccagacta tggtgccctc tagtgctatg gtgcctctgg cccagccacc tgctccagcc
600
cctgtgctga ccccaggacc accccagtca ctgagcgctc cagtgcccaa gtctacacag
660
gccggcgagg ggactctgag tgaagctctg ctgcacctgc agttcgacgc tgatgaggac
720
ctgggagctc tgctggggaa cagcaccgat cccggagtgt tcacagatct ggcctccgtg
780
gacaactctg agtttcagca gctgctgaat cagggcgtgt ccatgtctca tagtacagcc
840
gaaccaatgc tgatggagta ccccgaagcc attacccggc tggtgaccgg cagccagcgg
900
ccccccgacc ccgctccaac tcccctggga accagcggcc tgcctaatgg gctgtccgga
960
gatgaagact tctcaagcat cgctgatatg gactttagtg ccctgctgtc acagatttcc
1020
tctagtgggc agggaggagg tggaagcggc ttcagcgtgg acaccagtgc cctgctggac
1080
ctgttcagcc cctcggtgac cgtgcccgac atgagcctgc ctgaccttga cagcagcctg
1140
gccagtatcc aagagctcct gtctccccag gagcccccca ggcctcccga ggcagagaac
1200
agcagcccgg attcagggaa gcagctggtg cactacacag cgcagccgct gttcctgctg
1260
gaccccggct ccgtggacac cgggagcaac gacctgccgg tgctgtttga gctgggagag
1320
ggctcctact tctccgaagg ggacggcttc gccgaggacc ccaccatctc cctgctgaca
1380
ggctcggagc ctcccaaagc caaggacccc actgtctcc
1419
<210> 40
<211> 130
<212> БЕЛОК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 40
Met Ala Ser Asn Phe Thr Gln Phe Val Leu Val Asp Asn Gly Gly Thr
1 5 10 15
Gly Asp Val Thr Val Ala Pro Ser Asn Phe Ala Asn Gly Val Ala Glu
20 25 30
Trp Ile Ser Ser Asn Ser Arg Ser Gln Ala Tyr Lys Val Thr Cys Ser
35 40 45
Val Arg Gln Ser Ser Ala Gln Lys Arg Lys Tyr Thr Ile Lys Val Glu
50 55 60
Val Pro Lys Val Ala Thr Gln Thr Val Gly Gly Val Glu Leu Pro Val
65 70 75 80
Ala Ala Trp Arg Ser Tyr Leu Asn Met Glu Leu Thr Ile Pro Ile Phe
85 90 95
Ala Thr Asn Ser Asp Cys Glu Leu Ile Val Lys Ala Met Gln Gly Leu
100 105 110
Leu Lys Asp Gly Asn Pro Ile Pro Ser Ala Ile Ala Ala Asn Ser Gly
115 120 125
Ile Tyr
130
<210> 41
<211> 390
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 41
atggcttcaa actttactca gttcgtgctc gtggacaatg gtgggacagg ggatgtgaca
60
gtggctcctt ctaatttcgc taatggggtg gcagagtgga tcagctccaa ctcacggagc
120
caggcctaca aggtgacatg cagcgtcagg cagtctagtg cccagaagag aaagtatacc
180
atcaaggtgg aggtccccaa agtggctacc cagacagtgg gcggagtcga actgcctgtc
240
gccgcttgga ggtcctacct gaacatggag ctcactatcc caattttcgc taccaattct
300
gactgtgaac tcatcgtgaa ggcaatgcag gggctcctca aagacggtaa tcctatccct
360
tccgccatcg ccgctaactc aggtatctac
390
<210> 42
<211> 6673
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 42
gtgcccgtca gtgggcagag cgcacatcgc ccacagtccc cgagaagttg gggggagggg
60
tcggcaattg aaccggtgcc tagagaaggt ggcgcggggt aaactgggaa agtgatgtcg
120
tgtactggct ccgccttttt cccgagggtg ggggagaacc gtatataagt gcagtagtcg
180
ccgtgaacgt tctttttcgc aacgggtttg ccgccagaac acaggtaagt gccgtgtgtg
240
gttcccgcgg gcctggcctc tttacgggtt atggcccttg cgtgccttga attacttcca
300
cctggctgca gtacgtgatt cttgatcccg agcttcgggt tggaagtggg tgggagagtt
360
cgaggccttg cgcttaagga gccccttcgc ctcgtgcttg agttgaggcc tggcctgggc
420
gctggggccg ccgcgtgcga atctggtggc accttcgcgc ctgtctcgct gctttcgata
480
agtctctagc catttaaaat ttttgatgac ctgctgcgac gctttttttc tggcaagata
540
gtcttgtaaa tgcgggccaa gatctgcaca ctggtatttc ggtttttggg gccgcgggcg
600
gcgacggggc ccgtgcgtcc cagcgcacat gttcggcgag gcggggcctg cgagcgcggc
660
caccgagaat cggacggggg tagtctcaag ctggccggcc tgctctggtg cctggcctcg
720
cgccgccgtg tatcgccccg ccctgggcgg caaggctggc ccggtcggca ccagttgcgt
780
gagcggaaag atggccgctt cccggccctg ctgcagggag ctcaaaatgg aggacgcggc
840
gctcgggaga gcgggcgggt gagtcaccca cacaaaggaa aagggccttt ccgtcctcag
900
ccgtcgcttc atgtgactcc acggagtacc gggcgccgtc caggcacctc gattagttct
960
cgagcttttg gagtacgtcg tctttaggtt ggggggaggg gttttatgcg atggagtttc
1020
cccacactga gtgggtggag actgaagtta ggccagcttg gcacttgatg taattctcct
1080
tggaatttgc cctttttgag tttggatctt ggttcattct caagcctcag acagtggttc
1140
aaagtttttt tcttccattt caggtgtcgt gacgtacggc caccatgaaa aggccggcgg
1200
ccacgaaaaa ggccggccag gcaaaaaaga aaaaggacaa gaagtacagc atcggcctgg
1260
ccatcggcac caactctgtg ggctgggccg tgatcaccga cgagtacaag gtgcccagca
1320
agaaattcaa ggtgctgggc aacaccgacc ggcacagcat caagaagaac ctgatcggag
1380
ccctgctgtt cgacagcggc gaaacagccg aggccacccg gctgaagaga accgccagaa
1440
gaagatacac cagacggaag aaccggatct gctatctgca agagatcttc agcaacgaga
1500
tggccaaggt ggacgacagc ttcttccaca gactggaaga gtccttcctg gtggaagagg
1560
ataagaagca cgagcggcac cccatcttcg gcaacatcgt ggacgaggtg gcctaccacg
1620
agaagtaccc caccatctac cacctgagaa agaaactggt ggacagcacc gacaaggccg
1680
acctgcggct gatctatctg gccctggccc acatgatcaa gttccggggc cacttcctga
1740
tcgagggcga cctgaacccc gacaacagcg acgtggacaa gctgttcatc cagctggtgc
1800
agacctacaa ccagctgttc gaggaaaacc ccatcaacgc cagcggcgtg gacgccaagg
1860
ccatcctgtc tgccagactg agcaagagca gacggctgga aaatctgatc gcccagctgc
1920
ccggcgagaa gaagaatggc ctgttcggca acctgattgc cctgagcctg ggcctgaccc
1980
ccaacttcaa gagcaacttc gacctggccg aggatgccaa actgcagctg agcaaggaca
2040
cctacgacga cgacctggac aacctgctgg cccagatcgg cgaccagtac gccgacctgt
2100
ttctggccgc caagaacctg tccgacgcca tcctgctgag cgacatcctg agagtgaaca
2160
ccgagatcac caaggccccc ctgagcgcct ctatgatcaa gagatacgac gagcaccacc
2220
aggacctgac cctgctgaaa gctctcgtgc ggcagcagct gcctgagaag tacaaagaga
2280
ttttcttcga ccagagcaag aacggctacg ccggctacat tgacggcgga gccagccagg
2340
aagagttcta caagttcatc aagcccatcc tggaaaagat ggacggcacc gaggaactgc
2400
tcgtgaagct gaacagagag gacctgctgc ggaagcagcg gaccttcgac aacggcagca
2460
tcccccacca gatccacctg ggagagctgc acgccattct gcggcggcag gaagattttt
2520
acccattcct gaaggacaac cgggaaaaga tcgagaagat cctgaccttc cgcatcccct
2580
actacgtggg ccctctggcc aggggaaaca gcagattcgc ctggatgacc agaaagagcg
2640
aggaaaccat caccccctgg aacttcgagg aagtggtgga caagggcgct tccgcccaga
2700
gcttcatcga gcggatgacc aacttcgata agaacctgcc caacgagaag gtgctgccca
2760
agcacagcct gctgtacgag tacttcaccg tgtataacga gctgaccaaa gtgaaatacg
2820
tgaccgaggg aatgagaaag cccgccttcc tgagcggcga gcagaaaaag gccatcgtgg
2880
acctgctgtt caagaccaac cggaaagtga ccgtgaagca gctgaaagag gactacttca
2940
agaaaatcga gtgcttcgac tccgtggaaa tctccggcgt ggaagatcgg ttcaacgcct
3000
ccctgggcac ataccacgat ctgctgaaaa ttatcaagga caaggacttc ctggacaatg
3060
aggaaaacga ggacattctg gaagatatcg tgctgaccct gacactgttt gaggacagag
3120
agatgatcga ggaacggctg aaaacctatg cccacctgtt cgacgacaaa gtgatgaagc
3180
agctgaagcg gcggagatac accggctggg gcaggctgag ccggaagctg atcaacggca
3240
tccgggacaa gcagtccggc aagacaatcc tggatttcct gaagtccgac ggcttcgcca
3300
acagaaactt catgcagctg atccacgacg acagcctgac ctttaaagag gacatccaga
3360
aagcccaggt gtccggccag ggcgatagcc tgcacgagca cattgccaat ctggccggca
3420
gccccgccat taagaagggc atcctgcaga cagtgaaggt ggtggacgag ctcgtgaaag
3480
tgatgggccg gcacaagccc gagaacatcg tgatcgaaat ggccagagag aaccagacca
3540
cccagaaggg acagaagaac agccgcgaga gaatgaagcg gatcgaagag ggcatcaaag
3600
agctgggcag ccagatcctg aaagaacacc ccgtggaaaa cacccagctg cagaacgaga
3660
agctgtacct gtactacctg cagaatgggc gggatatgta cgtggaccag gaactggaca
3720
tcaaccggct gtccgactac gatgtggacc acatcgtgcc tcagagcttt ctgaaggacg
3780
actccatcga caacaaggtg ctgaccagaa gcgacaaggc ccggggcaag agcgacaacg
3840
tgccctccga agaggtcgtg aagaagatga agaactactg gcggcagctg ctgaacgcca
3900
agctgattac ccagagaaag ttcgacaatc tgaccaaggc cgagagaggc ggcctgagcg
3960
aactggataa ggccggcttc atcaagagac agctggtgga aacccggcag atcacaaagc
4020
acgtggcaca gatcctggac tcccggatga acactaagta cgacgagaat gacaagctga
4080
tccgggaagt gaaagtgatc accctgaagt ccaagctggt gtccgatttc cggaaggatt
4140
tccagtttta caaagtgcgc gagatcaaca actaccacca cgcccacgac gcctacctga
4200
acgccgtcgt gggaaccgcc ctgatcaaaa agtaccctaa gctggaaagc gagttcgtgt
4260
acggcgacta caaggtgtac gacgtgcgga agatgatcgc caagagcgag caggaaatcg
4320
gcaaggctac cgccaagtac ttcttctaca gcaacatcat gaactttttc aagaccgaga
4380
ttaccctggc caacggcgag atccggaagc ggcctctgat cgagacaaac ggcgaaaccg
4440
gggagatcgt gtgggataag ggccgggatt ttgccaccgt gcggaaagtg ctgagcatgc
4500
cccaagtgaa tatcgtgaaa aagaccgagg tgcagacagg cggcttcagc aaagagtcta
4560
tcctgcccaa gaggaacagc gataagctga tcgccagaaa gaaggactgg gaccctaaga
4620
agtacggcgg cttcgacagc cccaccgtgg cctattctgt gctggtggtg gccaaagtgg
4680
aaaagggcaa gtccaagaaa ctgaagagtg tgaaagagct gctggggatc accatcatgg
4740
aaagaagcag cttcgagaag aatcccatcg actttctgga agccaagggc tacaaagaag
4800
tgaaaaagga cctgatcatc aagctgccta agtactccct gttcgagctg gaaaacggcc
4860
ggaagagaat gctggcctct gccggcgaac tgcagaaggg aaacgaactg gccctgccct
4920
ccaaatatgt gaacttcctg tacctggcca gccactatga gaagctgaag ggctcccccg
4980
aggataatga gcagaaacag ctgtttgtgg aacagcacaa gcactacctg gacgagatca
5040
tcgagcagat cagcgagttc tccaagagag tgatcctggc cgacgctaat ctggacaaag
5100
tgctgtccgc ctacaacaag caccgggata agcccatcag agagcaggcc gagaatatca
5160
tccacctgtt taccctgacc aatctgggag cccctgccgc cttcaagtac tttgacacca
5220
ccatcgaccg gaagaggtac accagcacca aagaggtgct ggacgccacc ctgatccacc
5280
agagcatcac cggcctgtac gagacacgga tcgacctgtc tcagctggga ggcgacagcg
5340
ctggaggagg tggaagcgga ggaggaggaa gcggaggagg aggtagcgga cctaagaaaa
5400
agaggaaggt ggcggccgct ggatccggac gggctgacgc attggacgat tttgatctgg
5460
atatgctggg aagtgacgcc ctcgatgatt ttgaccttga catgcttggt tcggatgccc
5520
ttgatgactt tgacctcgac atgctcggca gtgacgccct tgatgatttc gacctggaca
5580
tgctgattaa ctgtacaggc agtggagagg gcagaggaag tctgctaaca tgcggtgacg
5640
tcgaggagaa tcctggccca atggccaagc ctttgtctca agaagaatcc accctcattg
5700
aaagagcaac ggctacaatc aacagcatcc ccatctctga agactacagc gtcgccagcg
5760
cagctctctc tagcgacggc cgcatcttca ctggtgtcaa tgtatatcat tttactgggg
5820
gaccttgtgc agaactcgtg gtgctgggca ctgctgctgc tgcggcagct ggcaacctga
5880
cttgtatcgt cgcgatcgga aatgagaaca ggggcatctt gagcccctgc ggacggtgcc
5940
gacaggtgct tctcgatctg catcctggga tcaaagccat agtgaaggac agtgatggac
6000
agccgacggc agttgggatt cgtgaattgc tgccctctgg ttatgtgtgg gagggctaag
6060
aattcgatat caagcttatc ggtaatcaac ctctggatta caaaatttgt gaaagattga
6120
ctggtattct taactatgtt gctcctttta cgctatgtgg atacgctgct ttaatgcctt
6180
tgtatcatgc tattgcttcc cgtatggctt tcattttctc ctccttgtat aaatcctggt
6240
tgctgtctct ttatgaggag ttgtggcccg ttgtcaggca acgtggcgtg gtgtgcactg
6300
tgtttgctga cgcaaccccc actggttggg gcattgccac cacctgtcag ctcctttccg
6360
ggactttcgc tttccccctc cctattgcca cggcggaact catcgccgcc tgccttgccc
6420
gctgctggac aggggctcgg ctgttgggca ctgacaattc cgtggtgttg tcggggaaat
6480
catcgtcctt tccttggctg ctcgcctgtg ttgccacctg gattctgcgc gggacgtcct
6540
tctgctacgt cccttcggcc ctcaatccag cggaccttcc ttcccgcggc ctgctgccgg
6600
ctctgcggcc tcttccgcgt cttcgccttc gccctcagac gagtcggatc tccctttggg
6660
ccgcctcccc gca
6673
<210> 43
<211> 4324
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Синтетический
<400> 43
gtgcccgtca gtgggcagag cgcacatcgc ccacagtccc cgagaagttg gggggagggg
60
tcggcaattg aaccggtgcc tagagaaggt ggcgcggggt aaactgggaa agtgatgtcg
120
tgtactggct ccgccttttt cccgagggtg ggggagaacc gtatataagt gcagtagtcg
180
ccgtgaacgt tctttttcgc aacgggtttg ccgccagaac acaggtaagt gccgtgtgtg
240
gttcccgcgg gcctggcctc tttacgggtt atggcccttg cgtgccttga attacttcca
300
cctggctgca gtacgtgatt cttgatcccg agcttcgggt tggaagtggg tgggagagtt
360
cgaggccttg cgcttaagga gccccttcgc ctcgtgcttg agttgaggcc tggcctgggc
420
gctggggccg ccgcgtgcga atctggtggc accttcgcgc ctgtctcgct gctttcgata
480
agtctctagc catttaaaat ttttgatgac ctgctgcgac gctttttttc tggcaagata
540
gtcttgtaaa tgcgggccaa gatctgcaca ctggtatttc ggtttttggg gccgcgggcg
600
gcgacggggc ccgtgcgtcc cagcgcacat gttcggcgag gcggggcctg cgagcgcggc
660
caccgagaat cggacggggg tagtctcaag ctggccggcc tgctctggtg cctggcctcg
720
cgccgccgtg tatcgccccg ccctgggcgg caaggctggc ccggtcggca ccagttgcgt
780
gagcggaaag atggccgctt cccggccctg ctgcagggag ctcaaaatgg aggacgcggc
840
gctcgggaga gcgggcgggt gagtcaccca cacaaaggaa aagggccttt ccgtcctcag
900
ccgtcgcttc atgtgactcc acggagtacc gggcgccgtc caggcacctc gattagttct
960
cgagcttttg gagtacgtcg tctttaggtt ggggggaggg gttttatgcg atggagtttc
1020
cccacactga gtgggtggag actgaagtta ggccagcttg gcacttgatg taattctcct
1080
tggaatttgc cctttttgag tttggatctt ggttcattct caagcctcag acagtggttc
1140
aaagtttttt tcttccattt caggtgtcgt gacgtacggc caccatggct tcaaacttta
1200
ctcagttcgt gctcgtggac aatggtggga caggggatgt gacagtggct ccttctaatt
1260
tcgctaatgg ggtggcagag tggatcagct ccaactcacg gagccaggcc tacaaggtga
1320
catgcagcgt caggcagtct agtgcccaga agagaaagta taccatcaag gtggaggtcc
1380
ccaaagtggc tacccagaca gtgggcggag tcgaactgcc tgtcgccgct tggaggtcct
1440
acctgaacat ggagctcact atcccaattt tcgctaccaa ttctgactgt gaactcatcg
1500
tgaaggcaat gcaggggctc ctcaaagacg gtaatcctat cccttccgcc atcgccgcta
1560
actcaggtat ctacagcgct ggaggaggtg gaagcggagg aggaggaagc ggaggaggag
1620
gtagcggacc taagaaaaag aggaaggtgg cggccgctgg atccccttca gggcagatca
1680
gcaaccaggc cctggctctg gcccctagct ccgctccagt gctggcccag actatggtgc
1740
cctctagtgc tatggtgcct ctggcccagc cacctgctcc agcccctgtg ctgaccccag
1800
gaccacccca gtcactgagc gctccagtgc ccaagtctac acaggccggc gaggggactc
1860
tgagtgaagc tctgctgcac ctgcagttcg acgctgatga ggacctggga gctctgctgg
1920
ggaacagcac cgatcccgga gtgttcacag atctggcctc cgtggacaac tctgagtttc
1980
agcagctgct gaatcagggc gtgtccatgt ctcatagtac agccgaacca atgctgatgg
2040
agtaccccga agccattacc cggctggtga ccggcagcca gcggcccccc gaccccgctc
2100
caactcccct gggaaccagc ggcctgccta atgggctgtc cggagatgaa gacttctcaa
2160
gcatcgctga tatggacttt agtgccctgc tgtcacagat ttcctctagt gggcagggag
2220
gaggtggaag cggcttcagc gtggacacca gtgccctgct ggacctgttc agcccctcgg
2280
tgaccgtgcc cgacatgagc ctgcctgacc ttgacagcag cctggccagt atccaagagc
2340
tcctgtctcc ccaggagccc cccaggcctc ccgaggcaga gaacagcagc ccggattcag
2400
ggaagcagct ggtgcactac acagcgcagc cgctgttcct gctggacccc ggctccgtgg
2460
acaccgggag caacgacctg ccggtgctgt ttgagctggg agagggctcc tacttctccg
2520
aaggggacgg cttcgccgag gaccccacca tctccctgct gacaggctcg gagcctccca
2580
aagccaagga ccccactgtc tcctgtacag gcagtggaga gggcagagga agtctgctaa
2640
catgcggtga cgtcgaggag aatcctggcc caaccatgaa aaagcctgaa ctcaccgcta
2700
cctctgtcga gaagtttctg atcgaaaagt tcgacagcgt ctccgacctg atgcagctct
2760
ccgagggcga agaatctcgg gctttcagct tcgatgtggg agggcgtgga tatgtcctgc
2820
gggtgaatag ctgcgccgat ggtttctaca aagatcgcta tgtttatcgg cactttgcat
2880
ccgccgctct ccctattccc gaagtgcttg acattgggga gttcagcgag agcctgacct
2940
attgcatctc ccgccgtgca cagggtgtca ccttgcaaga cctgcctgaa accgaactgc
3000
ccgctgttct ccagcccgtc gccgaggcca tggatgccat cgctgccgcc gatcttagcc
3060
agaccagcgg gttcggccca ttcggacctc aaggaatcgg tcaatacact acatggcgcg
3120
atttcatctg cgctattgct gatccccatg tgtatcactg gcaaactgtg atggacgaca
3180
ccgtcagtgc ctccgtcgcc caggctctcg atgagctgat gctttgggcc gaggactgcc
3240
ccgaagtccg gcacctcgtg cacgccgatt tcggctccaa caatgtcctg accgacaatg
3300
gccgcataac agccgtcatt gactggagcg aggccatgtt cggggattcc caatacgagg
3360
tcgccaacat cttcttctgg aggccctggt tggcttgtat ggagcagcag acccgctact
3420
tcgagcggag gcatcccgag cttgcaggat ctcctcggct ccgggcttat atgctccgca
3480
ttggtcttga ccaactctat cagagcttgg ttgacggcaa tttcgatgat gcagcttggg
3540
ctcagggtcg ctgcgacgca atcgtccggt ccggagccgg gactgtcggg cgtacacaaa
3600
tcgcccgcag aagcgctgcc gtctggaccg atggctgtgt ggaagtgctc gccgatagtg
3660
gaaacagacg ccccagcact cgtcctaggg caaaggatct gcagtaatga gaattcgata
3720
tcaagcttat cggtaatcaa cctctggatt acaaaatttg tgaaagattg actggtattc
3780
ttaactatgt tgctcctttt acgctatgtg gatacgctgc tttaatgcct ttgtatcatg
3840
ctattgcttc ccgtatggct ttcattttct cctccttgta taaatcctgg ttgctgtctc
3900
tttatgagga gttgtggccc gttgtcaggc aacgtggcgt ggtgtgcact gtgtttgctg
3960
acgcaacccc cactggttgg ggcattgcca ccacctgtca gctcctttcc gggactttcg
4020
ctttccccct ccctattgcc acggcggaac tcatcgccgc ctgccttgcc cgctgctgga
4080
caggggctcg gctgttgggc actgacaatt ccgtggtgtt gtcggggaaa tcatcgtcct
4140
ttccttggct gctcgcctgt gttgccacct ggattctgcg cgggacgtcc ttctgctacg
4200
tcccttcggc cctcaatcca gcggaccttc cttcccgcgg cctgctgccg gctctgcggc
4260
ctcttccgcg tcttcgcctt cgccctcaga cgagtcggat ctccctttgg gccgcctccc
4320
cgca
4324
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СКРИНИНГОВАЯ ПЛАТФОРМА CRISPR/CAS ДЛЯ ИДЕНТИФИКАЦИИ ГЕНЕТИЧЕСКИХ МОДИФИКАТОРОВ ЗАСЕВА ИЛИ АГРЕГАЦИИ ТАУ-БЕЛКА | 2020 |
|
RU2819350C2 |
| ЭКСПРЕССИРУЮЩИЕ CAS МЫШИНЫЕ ЭМБРИОНАЛЬНЫЕ СТВОЛОВЫЕ КЛЕТКИ И МЫШИ И ИХ ПРИМЕНЕНИЯ | 2018 |
|
RU2782358C2 |
| Животные, отличные от человека, содержащие гуманизированный локус альбумина | 2020 |
|
RU2815514C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2777988C2 |
| МЫШИ, СОДЕРЖАЩИЕ МУТАЦИИ, ВСЛЕДСТВИЕ КОТОРЫХ ЭКСПРЕССИРУЕТСЯ УКОРОЧЕННЫЙ НА С-КОНЦЕ ФИБРИЛЛИН-1 | 2017 |
|
RU2721125C1 |
| НОВЫЕ ФЕРМЕНТЫ CRISPR И СИСТЕМЫ | 2016 |
|
RU2771826C2 |
| Средство разрезания двунитевой ДНК с помощью Cas12d белка из Katanobacteria и гибридной РНК, полученной путем слияния направляющей CRISPR РНК и scout РНК | 2020 |
|
RU2771626C1 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2737537C2 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ DEMEQUINA SEDIMINICOLA | 2019 |
|
RU2722933C1 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2023 |
|
RU2832308C2 |
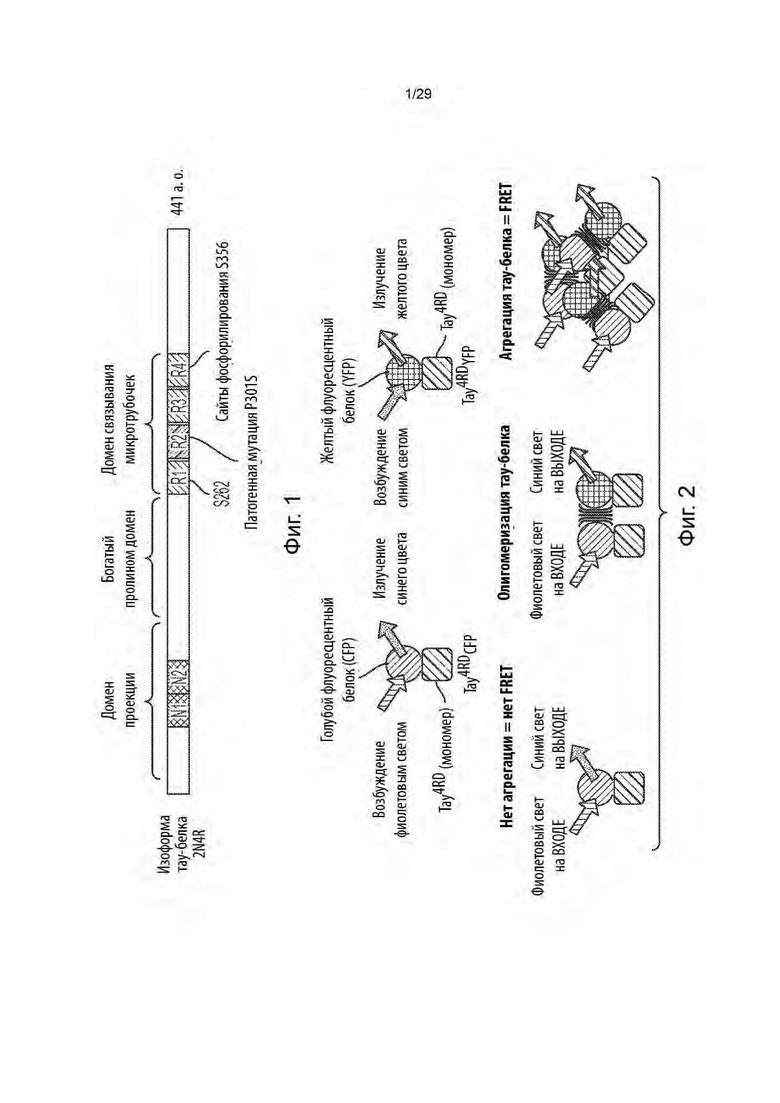
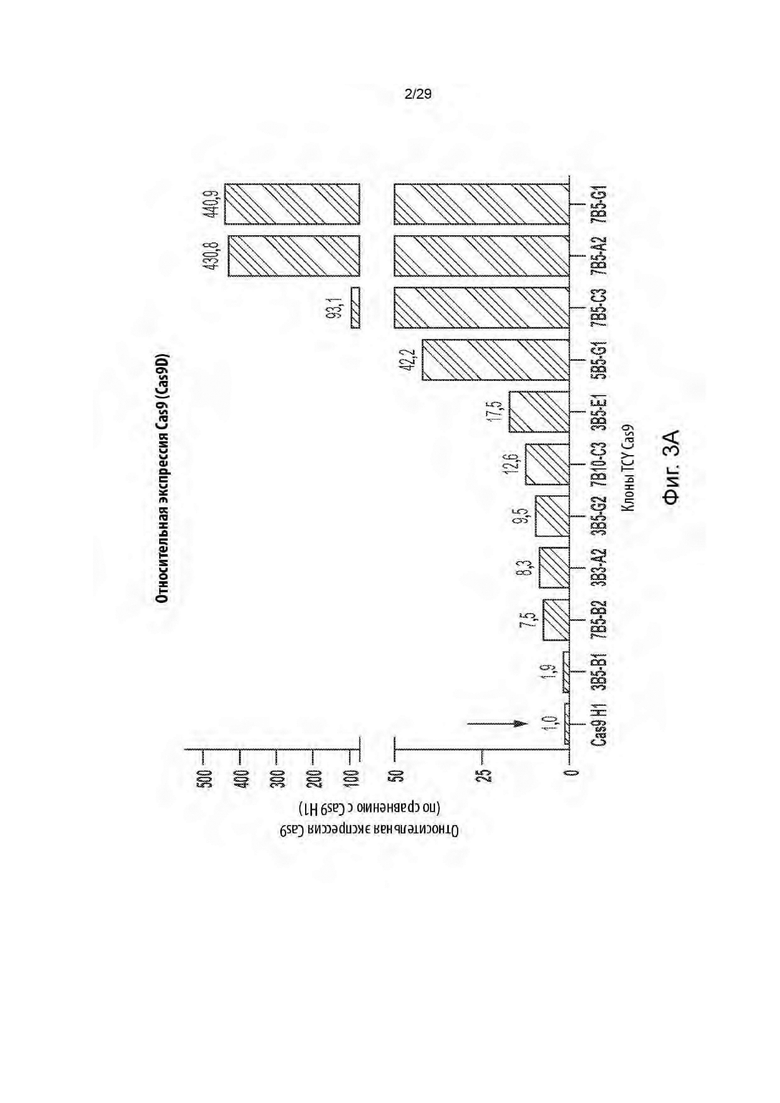
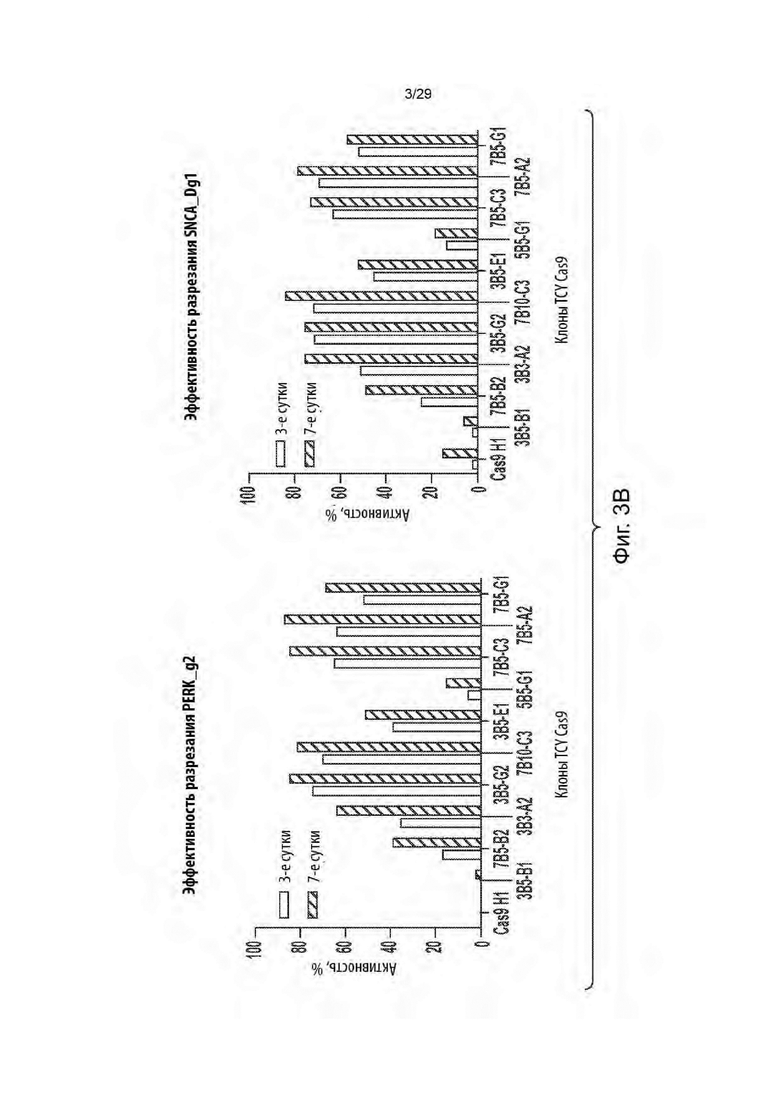
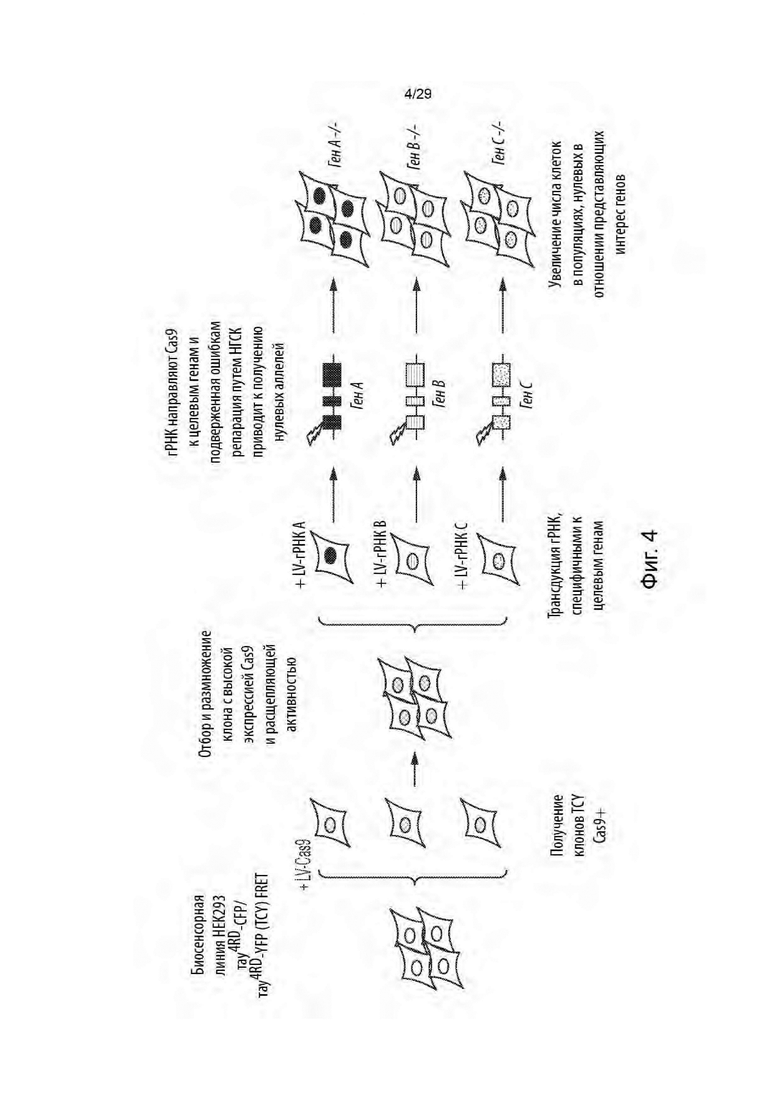
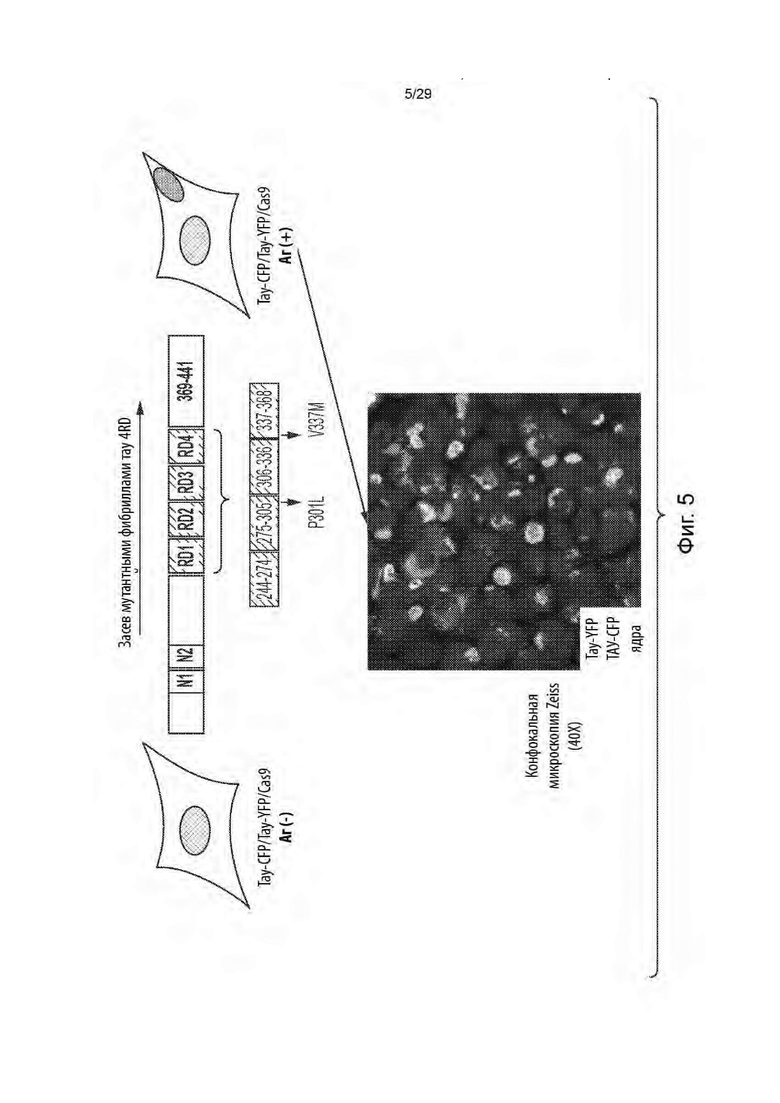
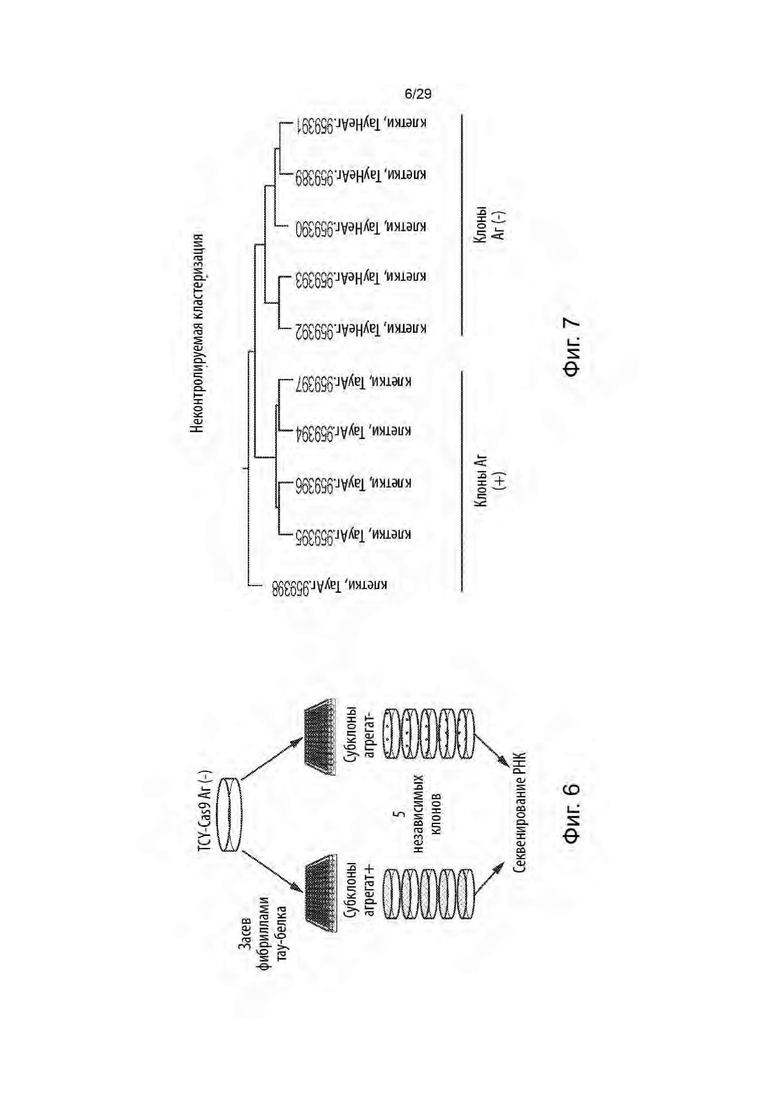
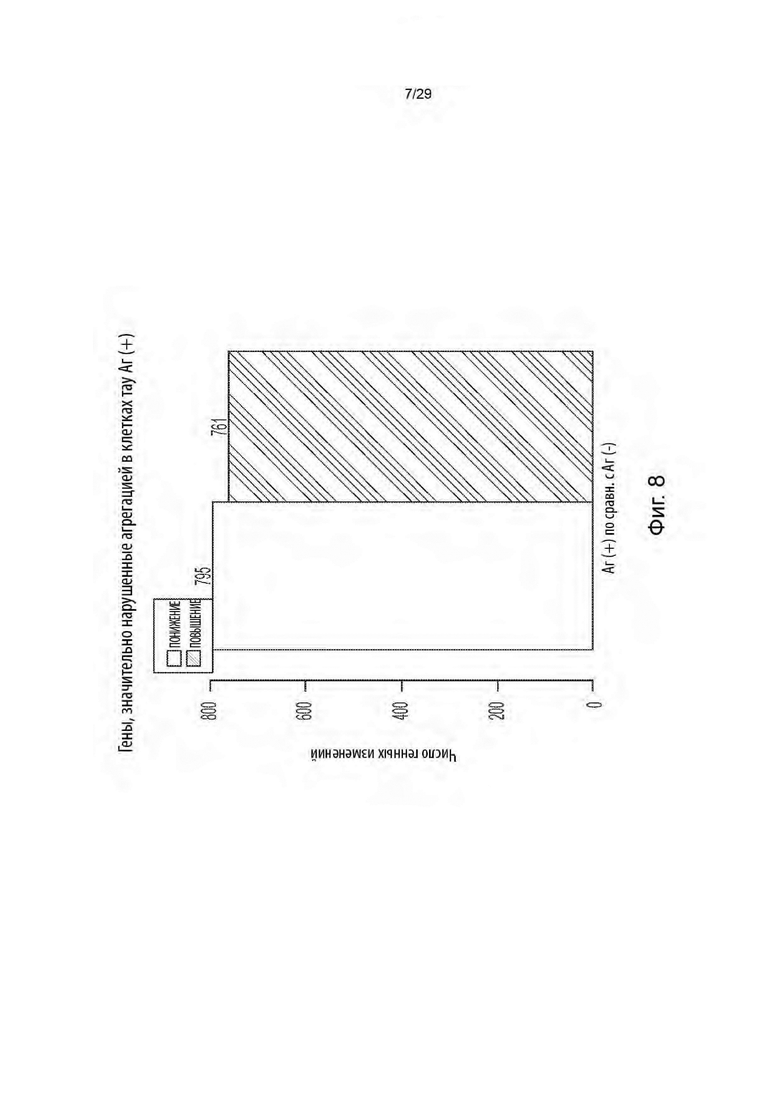
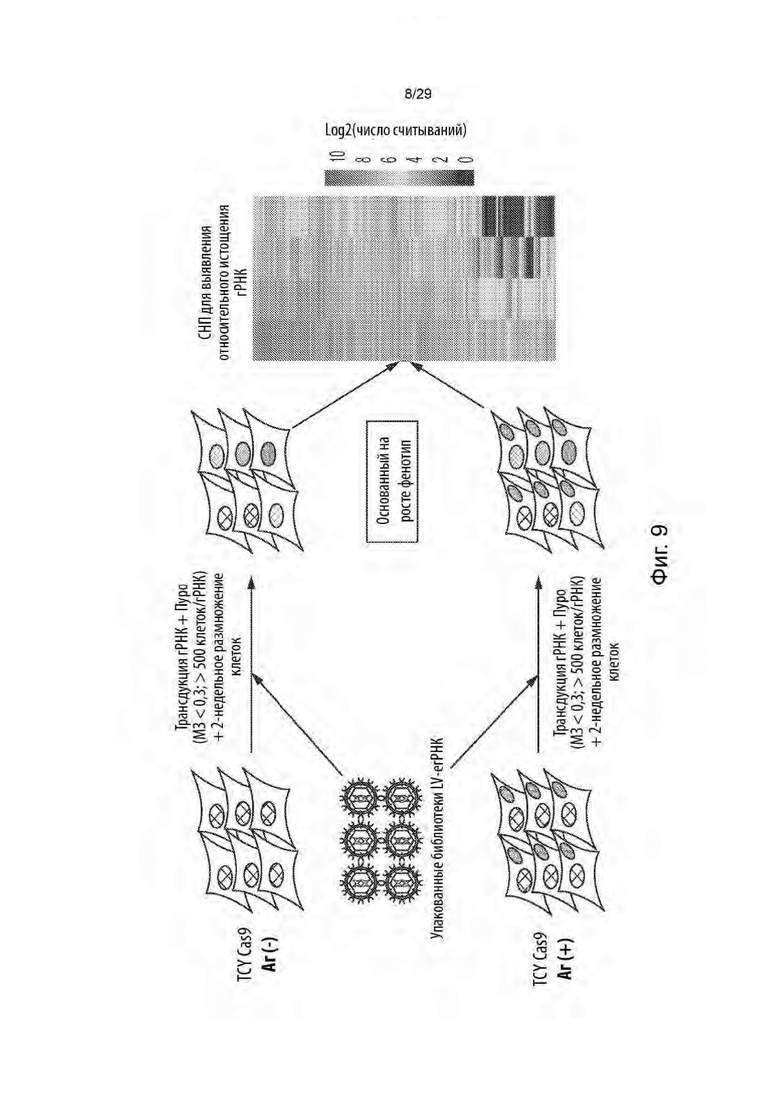
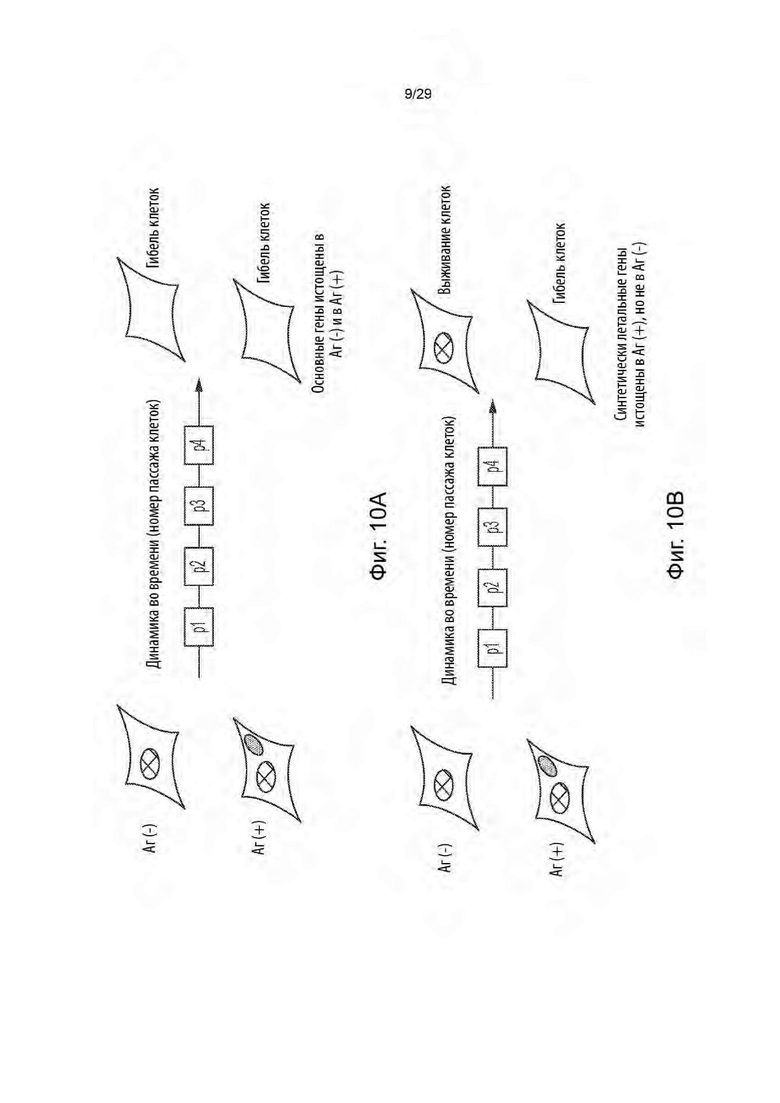
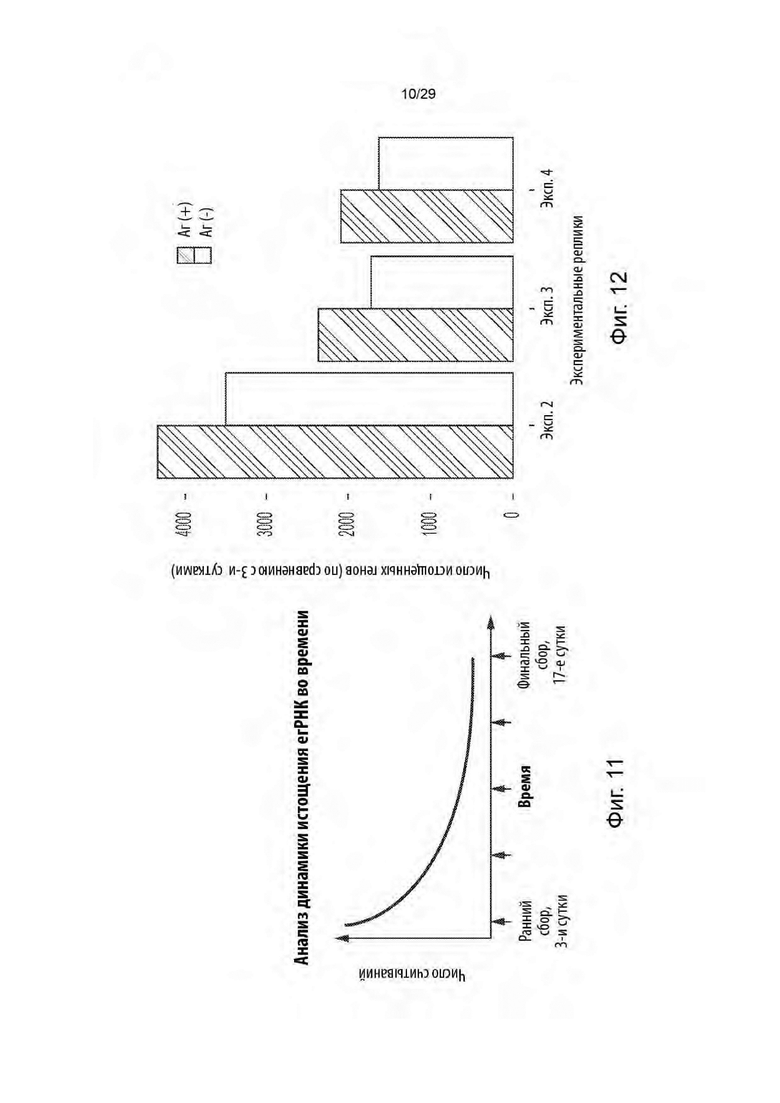
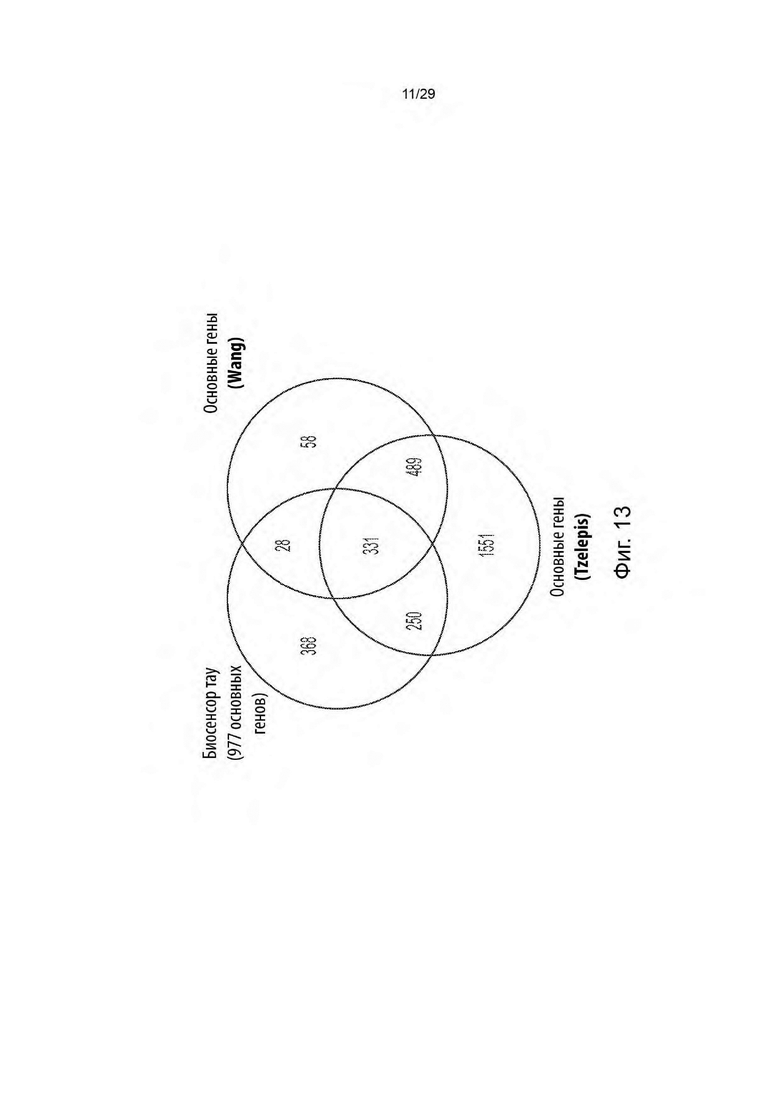
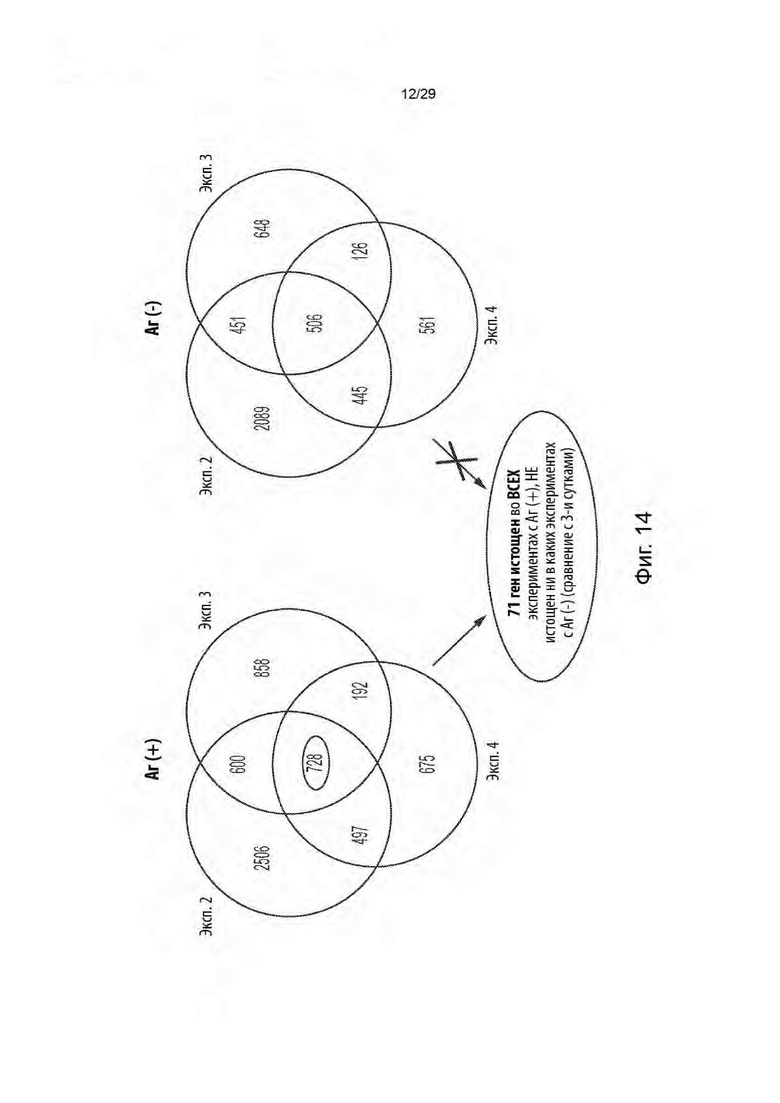
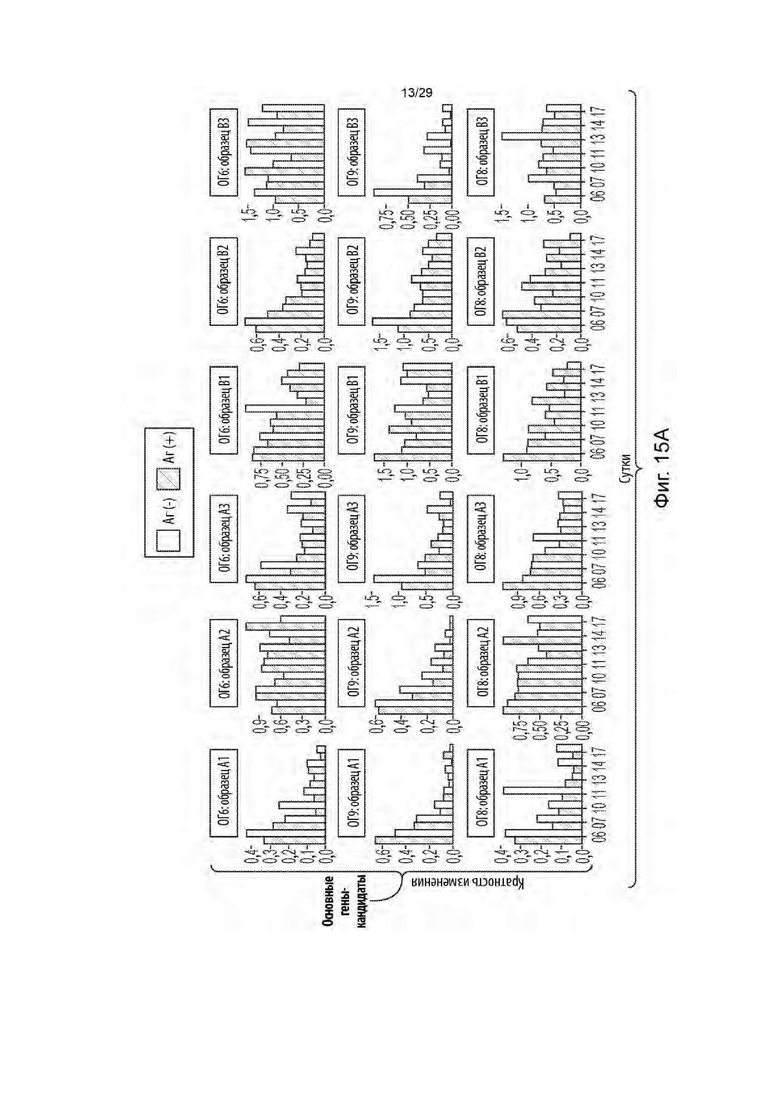
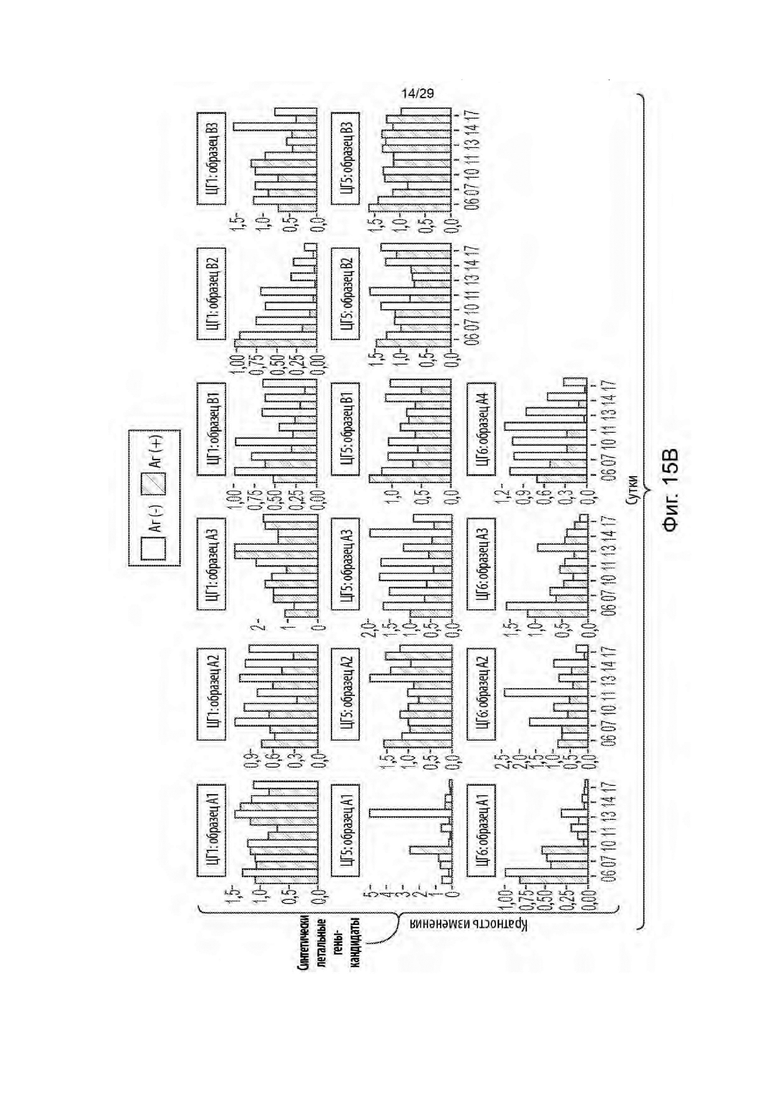
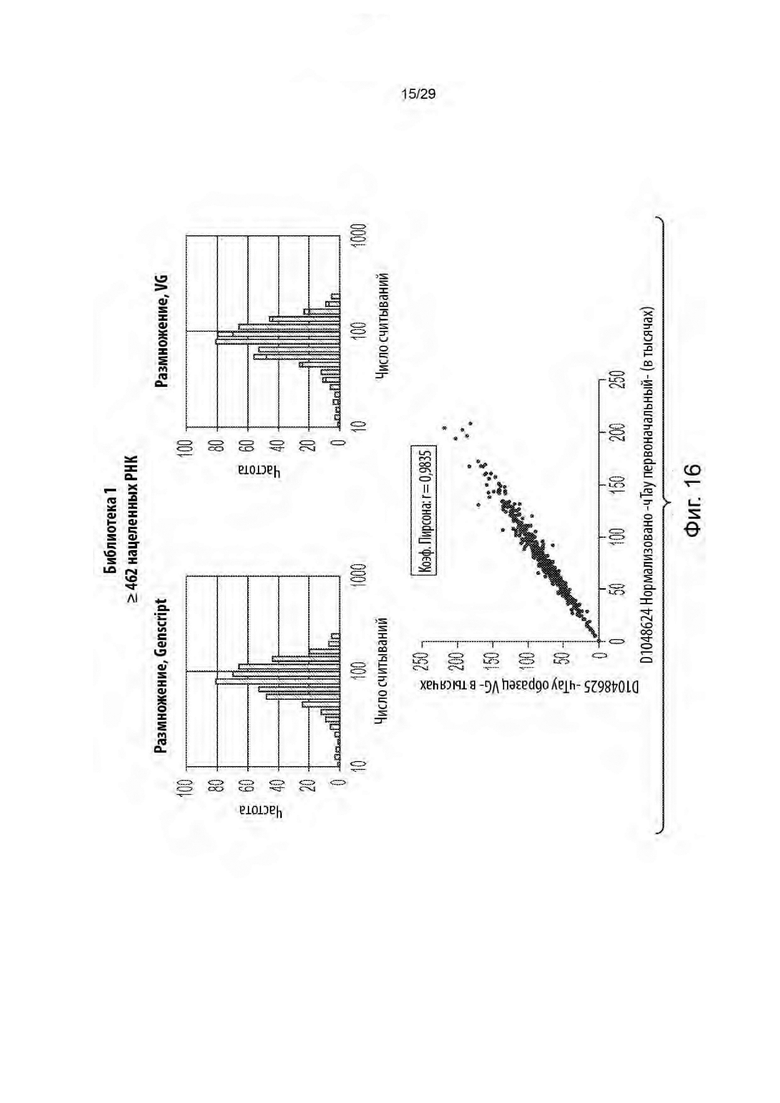
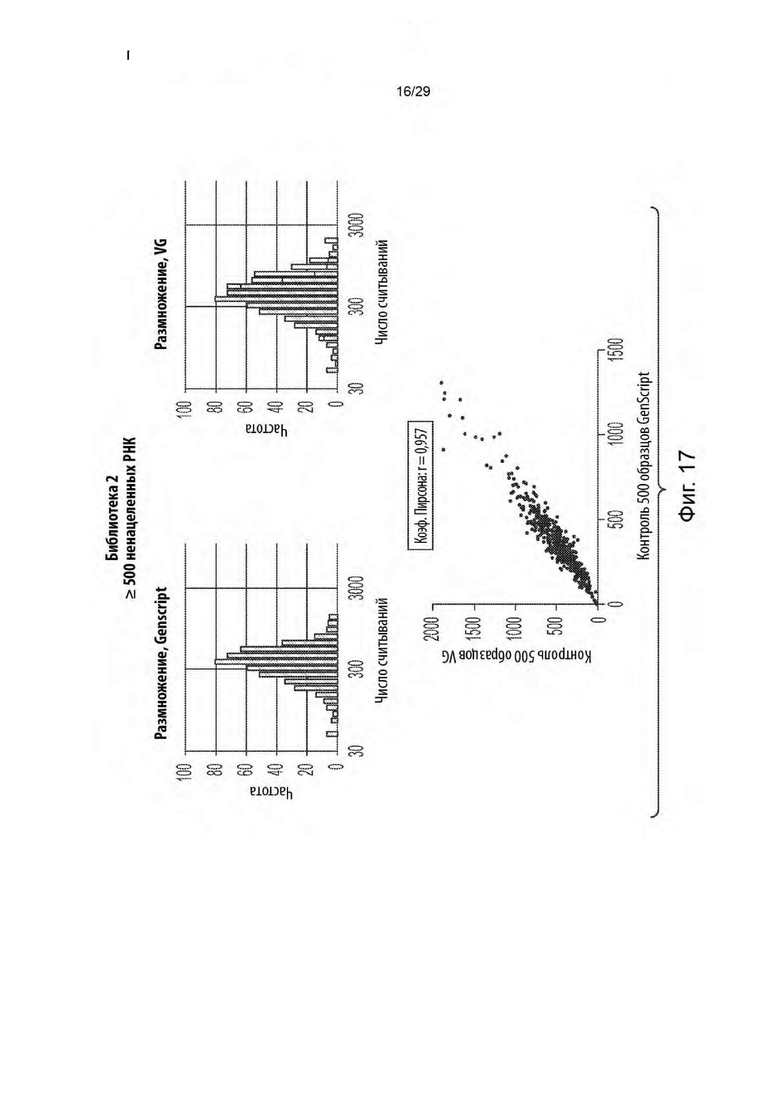
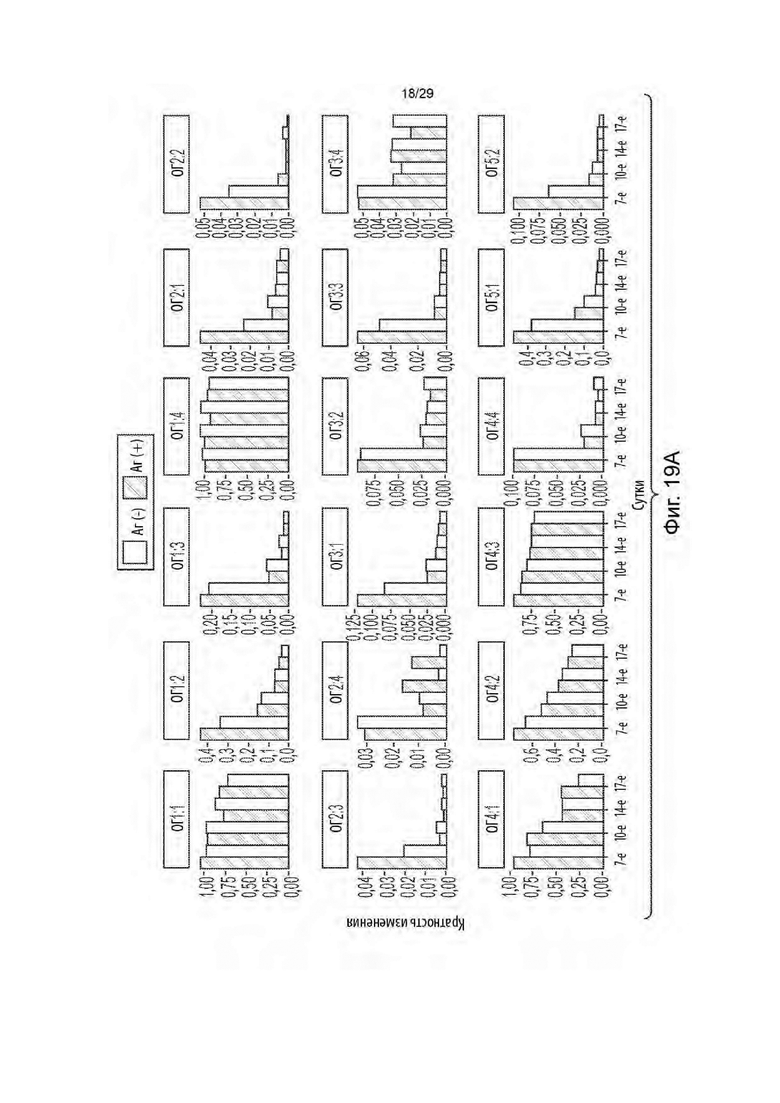
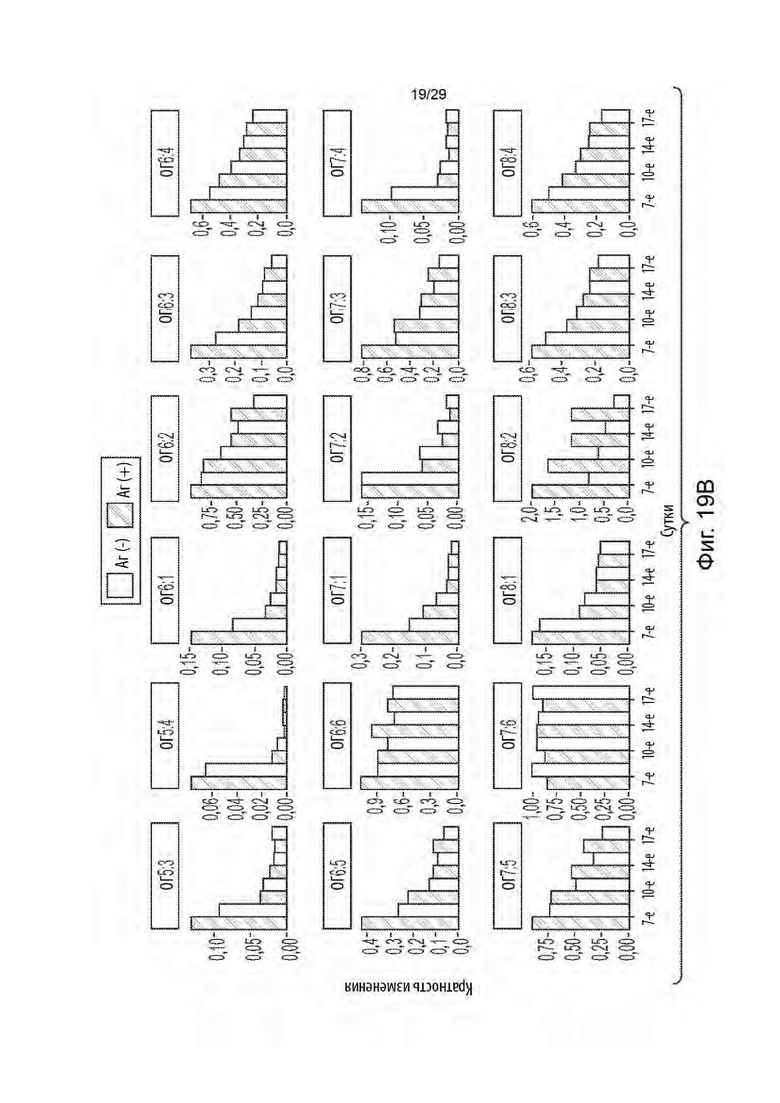
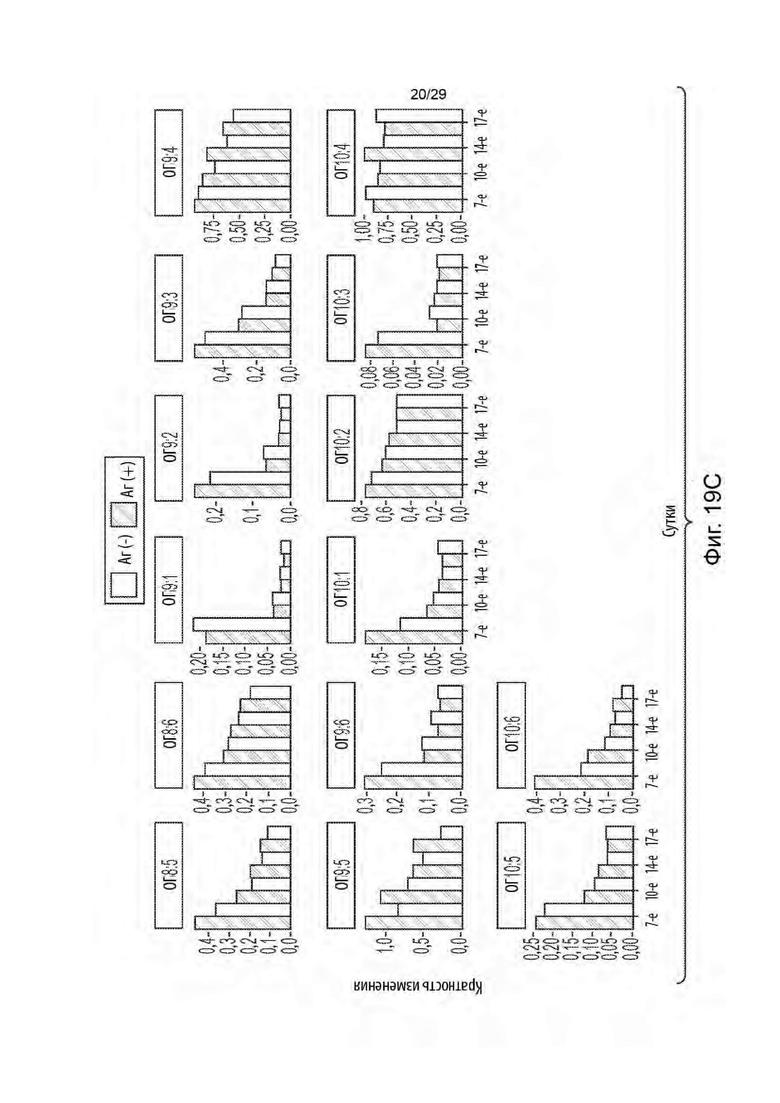
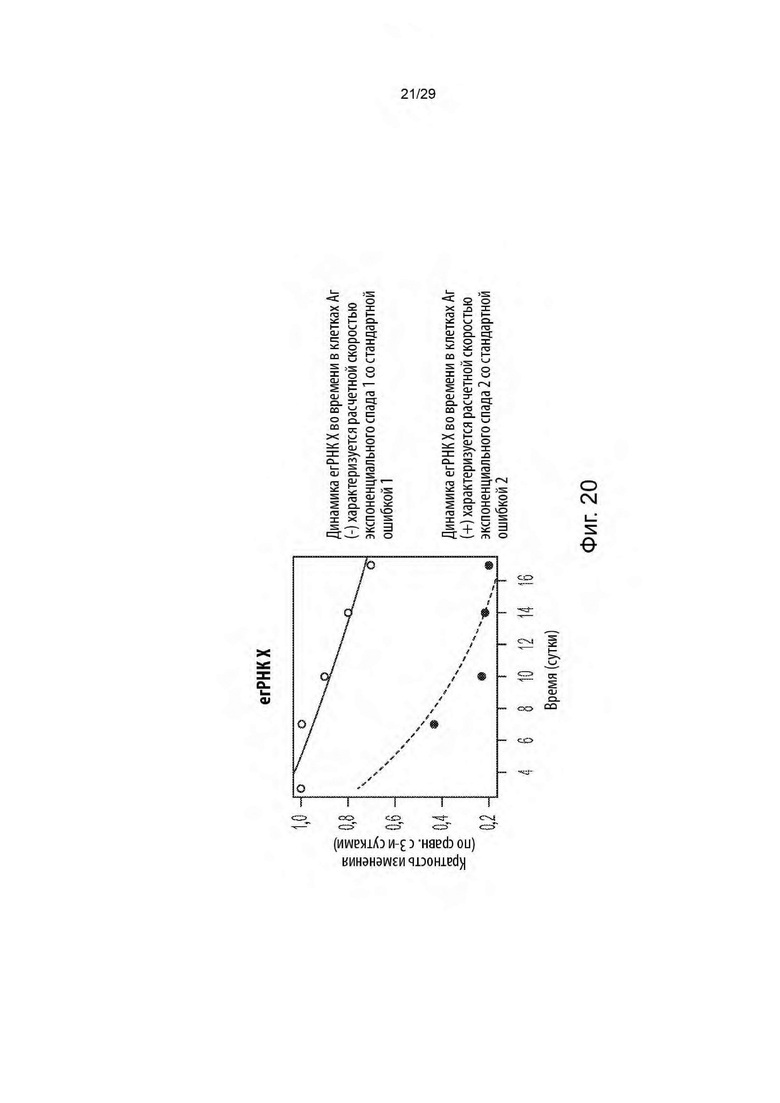
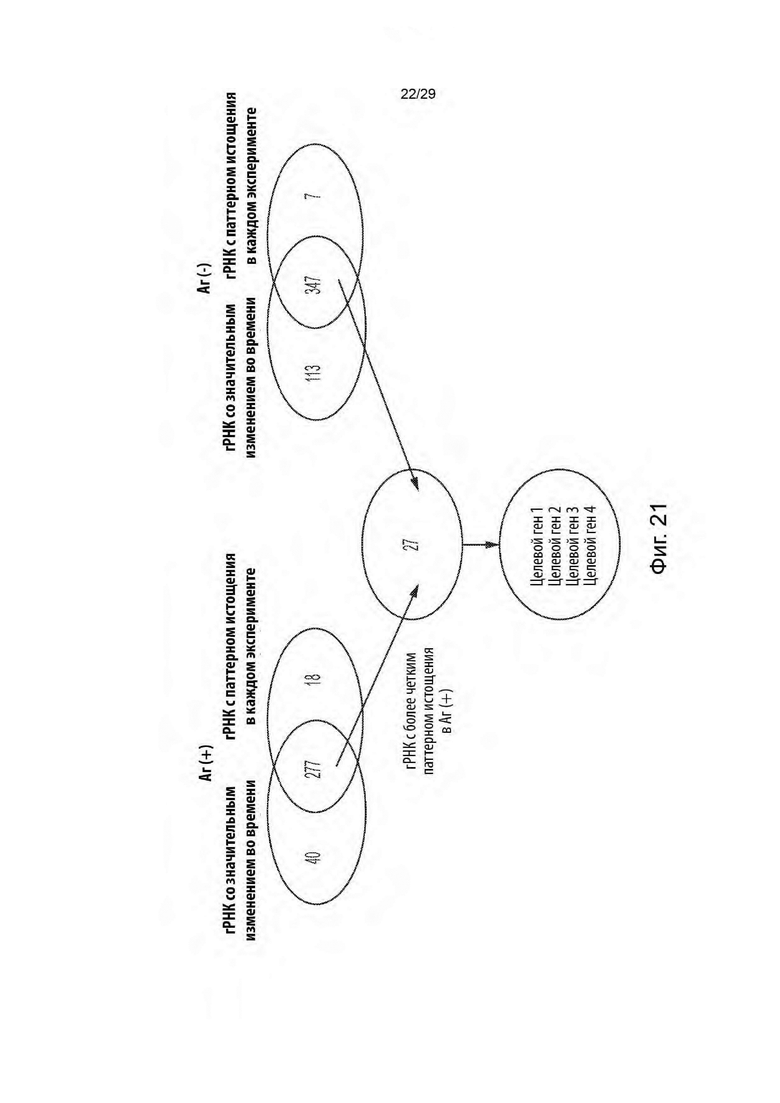
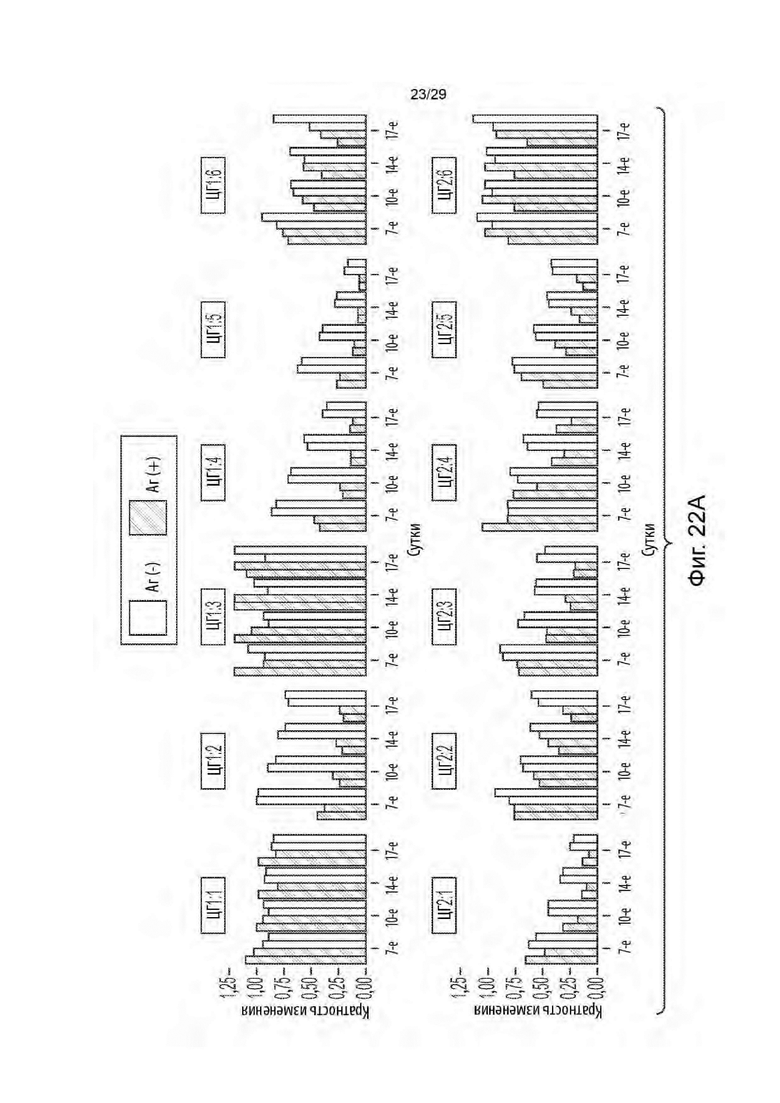

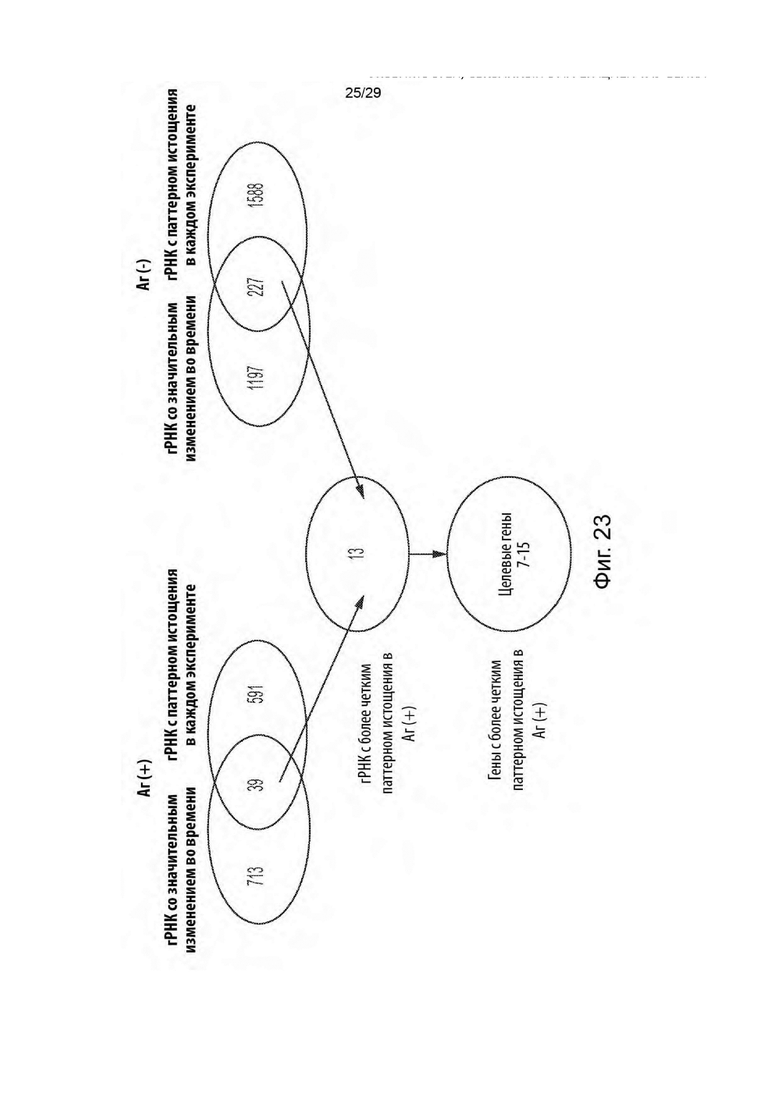

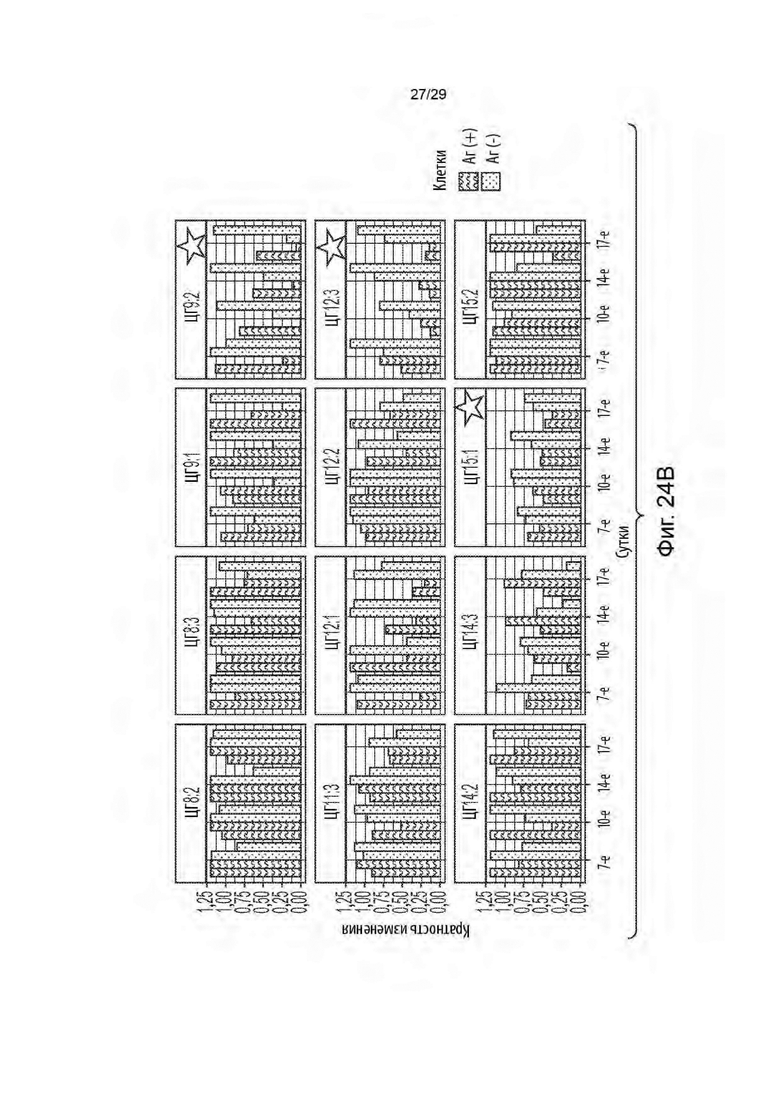
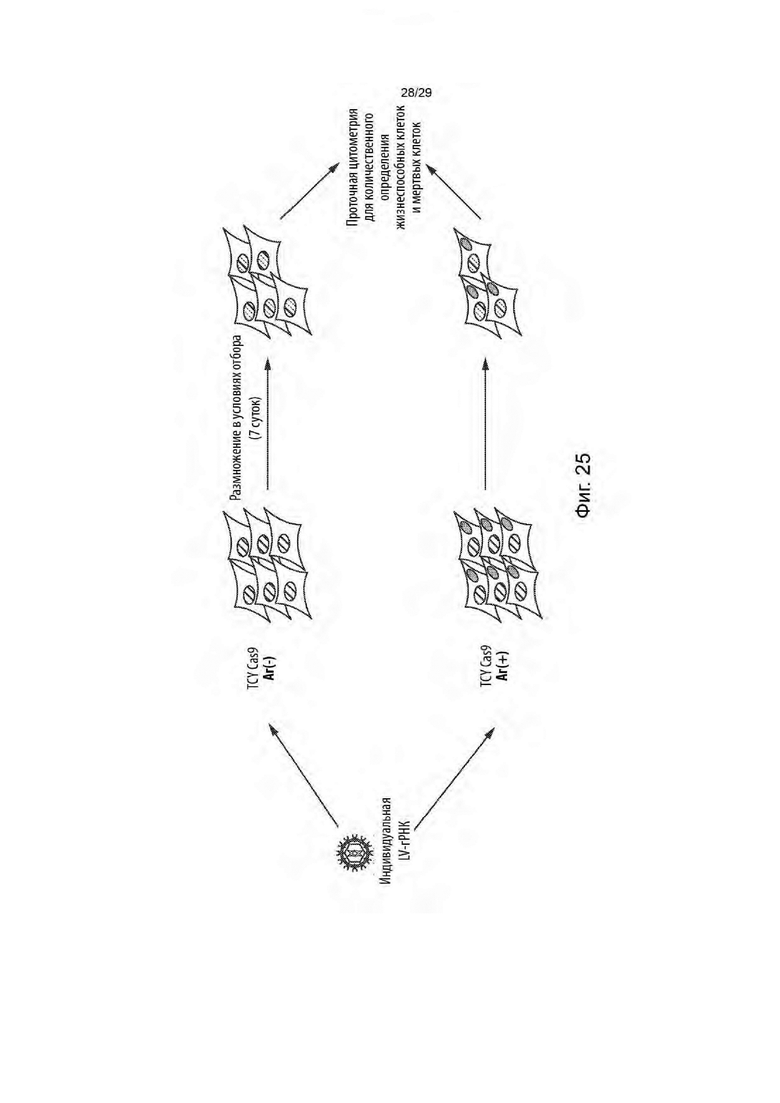

Изобретение относится к области биотехнологии, в частности к способам скрининга в отношении генетических уязвимостей, связанных с агрегацией тау-белка. Способы предусматривают обеспечение положительной в отношении агрегации популяции клеток и отрицательной в отношении агрегации популяции клеток, при этом каждая популяция клеток содержит белок Cas, введение в каждую популяцию клеток библиотеки, содержащей множество гидовых РНК, которые нацелены на множество генов, и определение содержания каждой из указанного множества гидовых РНК во множестве моментов времени. Изобретение эффективно для скрининга в отношении генетических уязвимостей, связанных с агрегацией тау-белка. 2 н. и 56 з.п. ф-лы, 26 ил., 5 табл., 4 пр.
1. Способ скрининга в отношении генетических уязвимостей, связанных с агрегацией тау-белка, включающий в себя:
(a) обеспечение положительной в отношении агрегации популяции клеток и отрицательной в отношении агрегации популяции клеток, при этом каждая популяция клеток содержит белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером,
где клетки представляют собой клетки млекопитающих,
где первый репортер и второй репортер представляют собой флуоресцентные белки и где первый репортер и второй репортер представляют собой пару с резонансным переносом энергии флуоресценции (FRET),
при этом указанная положительная в отношении агрегации популяция клеток содержит агрегаты первого содержащего повторы домена тау-белка, связанного с первым репортером, и второго содержащего повторы домена тау-белка, связанного со вторым репортером, и
при этом указанная отрицательная в отношении агрегации популяция клеток не содержит агрегатов первого содержащего повторы домена тау-белка, связанного с первым репортером, и второго содержащего повторы домена тау-белка, связанного со вторым репортером;
(b) введение в каждую популяцию клеток библиотеки, содержащей множество гидовых РНК, которые нацелены на множество генов, при этом указанное множество гидовых РНК образует комплексы с белком Cas и белок Cas расщепляет указанное множество генов, что приводит к нокауту генной функции; и
(c) определение содержания каждой из указанного множества гидовых РНК во множестве моментов времени в исследовании с течением времени в каждой популяции клеток,
где множество моментов времени включает в себя по меньшей мере три момента времени, при этом между каждым моментом времени проходит более 1 суток,
при этом истощение гидовой РНК в указанной положительной в отношении агрегации популяции клеток, но не в указанной отрицательной в отношении агрегации популяции клеток или большая скорость истощения гидовой РНК с течением времени в положительной в отношении агрегации популяции клеток по сравнению с отрицательной в отношении агрегации популяцией клеток указывает на то, что ген, на который нацелена данная гидовая РНК, проявляет синтетическую летальность с агрегатами тау-белка и является генетической уязвимостью, связанной с агрегацией тау-белка.
2. Способ по п. 1, отличающийся тем, что указанный белок Cas представляет собой белок Cas9.
3. Способ по п. 2, отличающийся тем, что указанный белок Cas представляет собой белок Cas9 из Streptococcus pyogenes.
4. Способ по п. 3, отличающийся тем, что указанный белок Cas содержит SEQ ID NO: 21, необязательно отличающийся тем, что указанный белок Cas кодируется кодирующей последовательностью, содержащей последовательность, указанную в SEQ ID NO: 22.
5. Способ по любому из предшествующих пунктов, отличающийся тем, что указанные белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, стабильно экспрессируются в указанных популяциях клеток.
6. Способ по любому из предшествующих пунктов, отличающийся тем, что нуклеиновые кислоты, кодирующие указанные белок Cas, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, геномно интегрированы в указанные популяции клеток.
7. Способ по любому из предшествующих пунктов, отличающийся тем, что каждая гидовая РНК нацелена на конститутивный экзон.
8. Способ по п. 7, отличающийся тем, что каждая гидовая РНК нацелена на конститутивный 5'-экзон.
9. Способ по любому из предшествующих пунктов, отличающийся тем, что каждая гидовая РНК нацелена на первый экзон, на второй экзон или на третий экзон.
10. Способ скрининга в отношении генетических уязвимостей, связанных с агрегацией тау-белка, включающий в себя:
(a) обеспечение положительной в отношении агрегации популяции клеток и отрицательной в отношении агрегации популяции клеток, при этом каждая популяция клеток содержит химерный белок Cas, который содержит белок Cas с инактивированной нуклеазной активностью, слитый с одним или большим числом доменов активации транскрипции, химерный адаптерный белок, который содержит адаптерный белок, слитый с одним или большим числом доменов активации транскрипции, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером,
где клетки представляют собой клетки млекопитающих,
где первый репортер и второй репортер представляют собой флуоресцентные белки и где первый репортер и второй репортер представляют собой пару с резонансным переносом энергии флуоресценции (FRET),
при этом указанная положительная в отношении агрегации популяция клеток содержит агрегаты первого содержащего повторы домена тау-белка, связанного с первым репортером, и второго содержащего повторы домена тау-белка, связанного со вторым репортером, и
при этом указанная отрицательная в отношении агрегации популяция клеток не содержит агрегаты первого содержащего повторы домена тау-белка, связанного с первым репортером, и второго содержащего повторы домена тау-белка, связанного со вторым репортером;
(b) введение в каждую популяцию клеток библиотеки, содержащей множество гидовых РНК, которые нацелены на множество генов, при этом указанное множество гидовых РНК образует комплексы с указанным химерным белком Cas и указанным химерным адаптерным белком и указанные комплексы активируют транскрипцию указанного множества генов, что приводит к повышенной генной экспрессии; и
(с) определение содержания каждой из указанного множества гидовых РНК во множестве моментов времени в исследовании с течением времени в каждой популяции клеток,
где множество моментов времени включает в себя по меньшей мере три момента времени, при этом между каждым моментом времени проходит более 1 суток,
при этом истощение гидовой РНК в указанной положительной в отношении агрегации популяции клеток, но не в указанной отрицательной в отношении агрегации популяции клеток или большая скорость истощения гидовой РНК с течением времени в положительной в отношении агрегации популяции клеток по сравнению с отрицательной в отношении агрегации популяцией клеток указывает на то, что активация гена, на который нацелена данная гидовая РНК, проявляет синтетическую летальность с агрегатами тау-белка и является генетической уязвимостью, связанной с агрегацией тау-белка.
11. Способ по п. 10, отличающийся тем, что указанный белок Cas представляет собой белок Cas9.
12. Способ по п. 11, отличающийся тем, что указанный белок Cas представляет собой белок Cas9 из Streptococcus pyogenes.
13. Способ по любому из пп. 10-12, отличающийся тем, что указанный химерный белок Cas содержит белок Cas с инактивированной нуклеазной активностью, слитый с доменом активации транскрипции VP64, необязательно отличающийся тем, что указанный химерный белок Cas содержит, в направлении от N-конца до С-конца: белок Cas с инактивированной нуклеазной активностью; сигнал ядерной локализации; и домен активатора транскрипции VP64.
14. Способ по любому из пп. 10-13, отличающийся тем, что указанный адаптерный белок представляет собой белок оболочки MS2, и отличающийся тем, что указанные один или большее число доменов активации транскрипции в указанном химерном адаптерном белке содержат домен активации транскрипции р65 и домен активации транскрипции HSF1, необязательно отличающийся тем, что указанный химерный адаптерный белок содержит, в направлении от N-конца до С-конца: белок оболочки MS2; сигнал ядерной локализации; домен активации транскрипции р65; и домен активации транскрипции HSF1.
15. Способ по любому из пп. 10-14, отличающийся тем, что указанный химерный белок Cas содержит SEQ ID NO: 36, необязательно отличающийся тем, что указанный химерный белок Cas кодируется кодирующей последовательностью, содержащей последовательность, указанную в SEQ ID NO: 38.
16. Способ по любому из пп. 10-15, отличающийся тем, что указанный химерный адаптерный белок содержит SEQ ID NO: 37, необязательно отличающийся тем, что указанный химерный адаптерный белок кодируется кодирующей последовательностью, содержащей последовательность, указанную в SEQ ID NO: 39.
17. Способ по любому из пп. 10-16, отличающийся тем, что указанный химерный белок Cas, химерный адаптерный белок, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, стабильно экспрессируются в популяции клеток.
18. Способ по любому из пп. 10-17, отличающийся тем, что нуклеиновые кислоты, кодирующие указанные химерный белок Cas, химерный адаптерный белок, первый содержащий повторы домен тау-белка, связанный с первым репортером, и второй содержащий повторы домен тау-белка, связанный со вторым репортером, геномно интегрированы в популяцию клеток.
19. Способ по любому из пп. 10-18, отличающийся тем, что каждая гидовая РНК нацелена на целевую последовательность для гидовой РНК в пределах 200 п. о. в направлении к 5' от сайта начала транскрипции.
20. Способ по любому из пп. 10-19, отличающийся тем, что каждая гидовая РНК содержит один или большее число адаптерсвязывающих элементов, с которыми может специфически связываться указанный химерный адаптерный белок, необязательно при этом каждая гидовая РНК содержит два адаптерсвязывающих элемента, с которыми может специфически связываться указанный химерный адаптерный белок, необязательно при этом первый адаптерсвязывающий элемент находится внутри первой петли каждой из одной или большего числа гидовых РНК, а второй адаптерсвязывающий элемент находится внутри второй петли каждой из одной или большего числа гидовых РНК, необязательно при этом указанный адаптерсвязывающий элемент содержит последовательность, указанную в SEQ ID NO: 33, и
необязательно при этом указанная каждая из одной или большего числа гидовых РНК представляет собой единую гидовую РНК, содержащую часть CRISPR-PHK (крРНК), слитую с частью трансактивирующей CRISPR-PHK (тракрРНК), и указанная первая петля представляет собой тетрапетлю, соответствующую остаткам 13-16 из SEQ ID NO: 17, а указанная вторая петля представляет собой шпильку 2, соответствующую остаткам 53-56 из SEQ ID NO: 17.
21. Способ по любому из предшествующих пунктов, отличающийся тем, что указанный первый содержащий повторы домен тау-белка и/или указанный второй содержащий повторы домен тау-белка представляют собой содержащий повторы домен тау-белка человека.
22. Способ по любому из предшествующих пунктов, отличающийся тем, что указанный первый содержащий повторы домен тау-белка и/или указанный второй содержащий повторы домен тау-белка содержат мутацию, способствующую агрегации.
23. Способ по п. 22, отличающийся тем, что указанный первый содержащий повторы домен тау-белка и/или указанный второй содержащий повторы домен тау-белка содержат мутацию тау-белка P301S.
24. Способ по любому из предшествующих пунктов, отличающийся тем, что указанный первый содержащий повторы домен тау-белка и/или указанный второй содержащий повторы домен тау-белка содержат домен тау-белка с четырьмя повторами.
25. Способ по любому из предшествующих пунктов, отличающийся тем, что указанный первый содержащий повторы домен тау-белка и/или указанный второй содержащий повторы домен тау-белка содержат SEQ ID NO: 11.
26. Способ по любому из предшествующих пунктов, отличающийся тем, что указанный первый содержащий повторы домен тау-белка и указанный второй содержащий повторы домен тау-белка являются одинаковыми.
27. Способ по любому из предшествующих пунктов, отличающийся тем, что указанный первый содержащий повторы домен тау-белка и указанный второй содержащий повторы домен тау-белка являются одинаковыми и каждый из них содержит домен тау-белка с четырьмя повторами, содержащий мутацию тау-белка P301S.
28. Способ по любому из предшествующих пунктов, отличающийся тем, что указанный первый репортер представляет собой голубой флуоресцентный белок (CFP), а указанный второй репортер представляет собой желтый флуоресцентный белок (YFP).
29. Способ по любому из предшествующих пунктов, отличающийся тем, что указанные клетки представляют собой клетки человека.
30. Способ по п. 29, отличающийся тем, что указанные клетки представляют собой клетки HEK293T.
31. Способ по любому из предшествующих пунктов, отличающийся тем, что указанное множество гидовых РНК вводят в концентрации, выбранной таким образом, чтобы большинство клеток получали только по одной из гидовых РНК.
32. Способ по любому из предшествующих пунктов, отличающийся тем, что указанное множество гидовых РНК нацелено на 100 или большее число генов, 1000 или большее число генов или 10000 или большее число генов.
33. Способ по любому из предшествующих пунктов, отличающийся тем, что указанная библиотека является полногеномной библиотекой.
34. Способ по любому из предшествующих пунктов, отличающийся тем, что нацеливание происходит в среднем на множество целевых последовательностей в каждом из целевого множества генов.
35. Способ по п. 34, отличающийся тем, что нацеливание происходит в среднем на по меньшей мере три целевые последовательности в каждом из целевого множества генов.
36. Способ по п. 35, отличающийся тем, что нацеливание происходит в среднем на от около трех до около шести целевых последовательностей в каждом из целевого множества генов.
37. Способ по любому из предшествующих пунктов, отличающийся тем, что указанное множество гидовых РНК вводят в указанные популяции клеток с помощью вирусной трансдукции.
38. Способ по п. 37, отличающийся тем, что каждое из указанного множества гидовых РНК находится в отдельном вирусном векторе.
39. Способ по п. 37 или 38, отличающийся тем, что указанное множество гидовых РНК вводят в указанные популяции клеток с помощью лентивирусной трансдукции.
40. Способ по любому из пп. 37-39, отличающийся тем, что указанные популяции клеток инфицированы с показателем множественности заражения, составляющим меньше чем около 0,3.
41. Способ по любому из предшествующих пунктов, отличающийся тем, что указанное множество гидовых РНК вводят в указанные популяции клеток вместе с маркером отбора и стадия (b) дополнительно включает в себя отбор клеток, которые содержат указанный маркер отбора.
42. Способ по п. 41, отличающийся тем, что указанный маркер отбора придает устойчивость к лекарственному средству, необязательно отличающийся тем, что указанный маркер отбора придает устойчивость к пуромицину или зеоцину.
43. Способ по п. 42, отличающийся тем, что указанный маркер отбора выбран из неомицинфосфотрансферазы, гигромицин-В-фосфотрансферазы, пуромицин-N-ацетилтрансферазы и бластицидин-S-дезаминазы.
44. Способ по любому из предшествующих пунктов, отличающийся тем, что каждая из указанных популяций клеток, в которые на стадии (b) вводят указанное множество гидовых РНК, включает в себя больше чем около 500 клеток на гидовую РНК.
45. Способ по любому из предшествующих пунктов, отличающийся тем, что указанное исследование с течением времени на стадии (с) имеет продолжительность больше чем около 1-й недели.
46. Способ по п. 45, отличающийся тем, что указанное исследование с течением времени на стадии (с) имеет продолжительность больше чем около 2 недель.
47. Способ по любому из предшествующих пунктов, отличающийся тем, что указанное исследование с течением времени на стадии (с) включает в себя от около 10 до около 15 удвоений клеток.
48. Способ по любому из предшествующих пунктов, отличающийся тем, что указанное множество моментов времени на стадии (с) включает в себя около четырех моментов времени или около шести моментов времени.
49. Способ по любому из предшествующих пунктов, отличающийся тем, что между каждым моментом времени на стадии (с) проходит более 2 суток.
50. Способ по п. 49, отличающийся тем, что между каждым моментом времени на стадии (с) проходит от около 3 суток до около 4 суток.
51. Способ по любому из предшествующих пунктов, отличающийся тем, что ген считается проявляющим синтетическую летальность с агрегатами тау-белка на стадии (с), если гидовая РНК, нацеленная на данный ген, истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток.
52. Способ по п. 51, отличающийся тем, что считается, что ген считается проявляющим синтетическую летальность с агрегатами тау-белка на стадии (с), если гидовая РНК, нацеленная на данный ген, имеет большую скорость истощения с течением времени в положительной в отношении агрегации популяции клеток по сравнению с отрицательной в отношении агрегации популяцией клеток.
53. Способ по любому из предшествующих пунктов, отличающийся тем, что гидовая РНК считается истощенной на стадии (с), если содержание данной гидовой РНК в каждый из моментов времени является меньшим или равным содержанию данной гидовой РНК в предшествующий момент времени.
54. Способ по любому из предшествующих пунктов, отличающийся тем, что гидовая РНК считается истощенной на стадии (с), если содержание данной гидовой РНК в каждый из моментов времени после второго момента времени является меньшим или равным содержанию в момент времени, на два момента времени предшествующий данному моменту времени.
55. Способ по любому из предшествующих пунктов, отличающийся тем, что ген считается проявляющим синтетическую летальность с агрегатами тау-белка на стадии (с), если больше чем около 30% гидовых РНК в библиотеке, нацеленных на данный ген, истощено в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток.
56. Способ по п. 55, отличающийся тем, что ген считается проявляющим синтетическую летальность с агрегатами тау-белка на стадии (с) в любой из следующих ситуаций:
(1) в библиотеке есть одна гидовая РНК, которая нацелена на данный ген, и данная одна гидовая РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток;
(2) в библиотеке есть две гидовые РНК, которые нацелены на данный ген, и по меньшей мере одна из данных двух гидовых РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток;
(3) в библиотеке есть три гидовые РНК, которые нацелены на данный ген, и по меньшей мере одна из данных трех гидовых РНК истощена в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток;
(4) в библиотеке есть четыре гидовые РНК, которые нацелены на данный ген, и по меньшей мере две из данных четырех гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток;
(5) в библиотеке есть пять гидовых РНК, которые нацелены на данный ген, и по меньшей мере две из данных пяти гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток; и
(6) в библиотеке есть шесть гидовых РНК, которые нацелены на данный ген, и по меньшей мере три из данных шести гидовых РНК истощены в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток.
57. Способ по любому из предшествующих пунктов, отличающийся тем, что указанный способ повторяют по меньшей мере три раза в по меньшей мере трех различных экспериментах, и ген считается проявляющим синтетическую летальность с агрегатами тау-белка, если данный ген считается проявляющим синтетическую летальность с агрегатами тау-белка в больше чем около 50% из указанных по меньшей мере трех различных экспериментов.
58. Способ по любому из предшествующих пунктов, отличающийся тем, что указанное исследование с течением времени на стадии (с) имеет продолжительность больше чем около 2 недель,
при этом указанное множество моментов времени на стадии (с) включает в себя около шести моментов времени,
при этом между каждым моментом времени на стадии (с) проходит от около 3 суток до около 4 суток,
при этом гидовая РНК считается истощенной на стадии (с), если содержание данной гидовой РНК в каждый из моментов времени после второго момента времени является меньшим или равным содержанию в момент времени, на два момента времени предшествующий данному моменту времени, и при этом ген считается проявляющим синтетическую летальность с агрегатами тау-белка на стадии (с), если больше чем около 30% гидовых РНК в библиотеке, нацеленных на данный ген, истощено в положительной в отношении агрегации популяции клеток, но не в отрицательной в отношении агрегации популяции клеток.
| Identification of genetic regulators for intracellular aggregation by genome-wide CRISPR screening, 2016 Fiscal Year Annual Research Report, The University of Tokyo, найдено в Интернет он-лайн по адресу https://kaken.nii.ac.jp/en/report/KAKENHI-PROJECT-16K15229/16K152292016jisseki/ | |||
| BRANDON B HOLMES et al., Proteopathic tau seeding predicts |
Авторы
Даты
2023-12-05—Публикация
2020-03-17—Подача