Область техники
Изобретение относится к области биотехнологии и молекулярной биологии, в частности генной инженерии, а именно к молекуле нуклеиновой кислоты для аллотопической экспрессии последовательности нуклеотидов, кодирующей белок МТ-ND4.
Уровень техники
Выяснение молекулярных механизмов развития наследственных заболеваний сетчатки (НЗС) обусловливает прогресс в разработке новых стратегий лечения этого заболевания [Scholl H.P.N, et al. Emerging therapies for inherited retinal degeneration // Sci. Transl. Med. American Association for the Advancement of Science, 2016. Vol.8, №368]. Существует специфический тип клеток нейрального происхождения, нарушение функционирования которых также обусловливает манифестацию НЗС - ганглиозные клетки сетчатки.
Первое клиническое описание пациента, страдающего наследственным заболеванием, проявляющимся в форме оптической нейропатии (наследственная оптическая нейропатия, Leber's Hereditary Optic Neuropathy, LHON) было сделано более 150 лет назад Альбрехтом фон Грефе. Позже в 1871 году заболевание было выделено в отдельную нозологическую единицу немецким офтальмологом Теодором Лебером [Leber Т. Ueber hereditare und congenital-angelegte Sehnervenleiden // Albr. von Graefe's Arch. für Ophthalmol. Springer, 1871. Vol.17, №2. P. 249-291]. Вследствие значительной половой диспропорции, а наследственная нейропатия зрительного нерва чаще проявляется у молодых мужчин, было показано, что тип наследования был материнским, а заболевание было вызвано мутациями в митохондриальном геноме, или мтДНК [Erickson R.P. Leber's optic atrophy, a possible example of maternal inheritance. // Am. J. Hum. Genet. Elsevier, 1972. Vol.24, №3. P. 348]. Дуглас Уоллес с коллегами в 1988 г. обнаружили первую мутацию митохондриальной ДНК (мтДНК), ассоциированную с LHON, в гене MT-ND4, кодирующем белок MT-ND4 - одну из субъединиц НАДН-дегидрогеназного комплекса электрон-транспортной цепи митохондрий [Brown M.D. et al. Functional Analysis of Lymphoblast and Cybrid Mitochondria Containing the 3460, 11778, or 14484 Leber's Hereditary Optic Neuropathy Mitochondrial DNA Mutation * // J. Biol. Chem. Elsevier, 2000. Vol.275, №51. P. 39831-39836]. Было описано множество других мутаций в генах, кодирующих компоненты НАДН-дегидрогеназного комплекса, которые участвуют в патогенезе LHON, однако мутации в гене MT-ND4 (G11778A) [Wallace D.C. et al. Mitochondrial DNA mutation associated with Leber's hereditary optic neuropathy //Science, 1988. Vol.242, №4884. P. 1427-1430], MT-ND1 (G3460A) и MT-ND6 (T14484C) [Huoponen K. et al. A new mtDNA mutation associated with Leber hereditary optic neuroretinopathy. // Am. J. Hum. Genet. Elsevier, 1991. Vol.48, №6. P. 1147] в совокупности ответственны за 90-95% всех случаев заболевания. При этом около 70% всех случаев связаны с мутацией в гене MT-ND4 [Yu-Wai-Man P., Griffiths P.G., Chinnery P. F. Mitochondrial optic neuropathies-disease mechanisms and therapeutic strategies //Progress in retinal and eye research. 2011. T. 30. №. 2. C. 81-114].
Эти замены вызывают острую или подострую гибель ганглиозных клеток сетчатки (retinal ganglion cells, RGC) и их аксонов, составляющих зрительный нерв, что приводит к потере центрального зрения и слепоте. Проявление патологии двустороннее: у большинства пациентов наблюдается потеря зрения сначала в одном глазу, а затем, как правило через 6-8 недель, - в другом. Лишь в 25% случаев происходит одновременное проявление нарушений в обоих глазах [Yu-Wai-Man P. et al. Treatment strategies for inherited optic neuropathies: past, present and future // Eye 2014 285. Nature Publishing Group, 2014. Vol.28, №5. P. 521-537].
Мутации, приводящие к LHON, нарушают работу комплекса I дыхательной цепи, а именно значительное снижают НАДН-убихинон-оксидоредуктазную активность НАДН-дегидрогеназного комплекса [Brown M.D. et al. Functional Analysis of Lymphoblast and Cybrid Mitochondria Containing the 3460, 11778, or 14484 Leber's Hereditary Optic Neuropathy Mitochondrial DNA Mutation * // J. Biol. Chem. Elsevier, 2000. Vol.275, №51. P. 39831-39836; Majander A. et al. Electron transfer properties of NADH: Ubiquinone reductase in the ND1/3460 and the ND4/11778 mutations of the Leber hereditary optic neuroretinopathy (LHON) // FEBS Lett. No longer published by Elsevier, 1991. Vol.292, №12. P. 289 292; Howell N. et al. Leber hereditary optic neuropathy: identification of the same mitochondrial ND1 mutation in six pedigrees. // Am. J. Hum. Genet. Elsevier, 1991. Vol.49, №5. P. 939; Smith P.R. et al. Platelet mitochondrial function in Leber's hereditary optic neuropathy // J. Neurol. Sci. Elsevier, 1994. Vol.122, №1. P. 80-83; Carelli V. et al. Leber's hereditary optic neuropathy // Neurology. Wolters Kluwer Health, Inc. on behalf of the American Academy of Neurology, 1997. Vol.48, №6. P. 1623-1632], что влечет гибель нейронов из-за истощения энергии в виде АТФ. Самые распространенные мутации находятся в генах MT-ND1, MT-ND6, MT-ND4, при этом на мутации в гене MT-ND4 приходится большинство случаев LHON [Majander A. et al. Electron transfer properties of NADH: Ubiquinone reductase in the ND1/3460 and the ND4/11778 mutations of the Leber hereditary optic neuroretinopathy (LHON) // FEBS Lett. No longer published by Elsevier, 1991. Vol.292, №1-2. P. 289-292; Carelli V. et al. Biochemical Features of mtDNA 14484 (ND6/M64V) Point Mutation Associated with Leber's Hereditary Optic Neuropathy. 1999; Hofhaus G. et al. Respiration and Growth Defects in Transmitochondrial Cell Lines Carrying the 11778 Mutation Associated with Leber's Hereditary Optic Neuropathy * // J. Biol. Chem. Elsevier, 1996. Vol.271, №22. P. 13155-13161].
Исследования клеточных культур цитоплазматических гибридов (цибридов) выявили снижение скорости роста мутантных клеток вместе с более низким потреблением кислорода. Группа Валерио Карелли показала, что энергетическая недостаточность определяет судьбу клеток в модели цитоплазматических гибридов LHON [Vergani L. et al. MtDNA Mutations Associated with Leber's Hereditary Optic Neuropathy: Studies on Cytoplasmic Hybrid (Cybrid) Cells // Biochem. Biophys. Res. Commun. Academic Press, 1995. Vol. 210, №3. P. 880-888]. Мутации комплекса I могут вызывать также окислительный стресс, потенциальной патогенетической роли которого уделялось меньше внимания.
Митохондриальное дыхание является основой метаболических процессов в клетках всех многоклеточных эукариот. Нарушение митохондриальных процессов, будь то фрагментация или другие морфологические изменения, повышенная частота мутаций в мтДНК, накопление мутантных белков, изменение проницаемости митохондриальных мембран, изменение окислительно-восстановительного потенциала и нарушение окислительного фосфорилирования, способны приводить к значительным последствиям для внутриклеточного метаболизма. В особенности, чувствительными к повреждению митохондрий и их дисфункции являются мышечные волокна и клетки нейрального происхождения. Окислительный стресс и повреждение митохондрий вовлечены в патогенез нескольких нейродегенеративных заболеваний, поскольку характеризуются перепроизводством активных форм кислорода, которые могут вызывать мутации митохондриальной ДНК, снижать эффективность митохондриальной дыхательной цепи, изменять проницаемость мембран и влиять на гомеостаз Са2+ и защитные системы митохондрий.
Дисфункция комплекса I дыхательной цепи, возникающая вследствие мутаций в мтДНК (m.11778G>A в MT-ND4; m.3460G>A в MT-ND1; m.14484Т>С в MT-ND6), приводит к снижению уровня синтеза АТФ. При вызванной подобной дисфункцией гипоксии гибнут наиболее чувствительные к повреждениям митохондрий ганглиозные клетки сетчатки. Локализация и функциональные особенности ганглиозных клеток сетчатки обусловливают их уникальные энергетические потребности: длинные аксоны этих клеток в ретробульбарной области переходят от немиелинизированного в миелинизированное состояние. Аксональный транспорт, обусловлен моторными белками кинезином и динеином, которым для выполнения этой функции требуется большое количество АТФ. Истощение энергии, происходящее при митохондриальной дисфункции, нарушает аксональный транспорт [Chinnery P.F. et al. Leber hereditary optic neuropathy: does heteroplasmy influence the inheritance and expression of the G11778A mitochondrial DNA mutation? //American journal of medical genetics. 2001. T. 98. №. 3. - C. 235-243]. Кроме того, апоптоз ганглиозных клеток сетчатки в конечном счете приводит к атрофии зрительного нерва.
Роль активных форм кислорода (АФК) в патогенезе LHON является дополнением к гипотезе дефицита АТФ, поскольку митохондрии являются местом окислительного фосфорилирования, а комплекс I дыхательной цепи - источником, в качестве побочных продуктов, большинства клеточных супероксидов (О2-) [Bolisetty S., Jaimes Е. А. Mitochondria and reactive oxygen species: physiology and pathophysiology //International journal of molecular sciences. 2013. T. 14. №. 3. C. 6306-6344], предшественников всех АФК [Juan С.A. et al. The chemistry of reactive oxygen species (ROS) revisited: outlining their role in biological macromolecules (DNA, lipids and proteins) and induced pathologies //International Journal of Molecular Sciences. - 2021. - T. 22. -№. 9. - C. 4642]. Хотя АФК играют внутриклеточную сигнальную роль в основных клеточных процессах [Starkov A.A. The role of mitochondria in reactive oxygen species metabolism and signaling //Annals of the New York Academy of Sciences. - 2008. - T. 1147. - №. 1. - C. 37-52], диапазон концентрации АФК с благоприятными физиологическими последствиями относительно узок.
В последние годы заместительная генная терапия рассматривалась как потенциальное решение проблемы лечения LHON. При заместительной генной терапии в клетку-мишень доставляют копию гена, не содержащую мутаций, которая обеспечивает синтез функционального белка. Однако генная терапия мутаций митохондриальной ДНК была сложной задачей, поскольку наличие двойной мембраны может препятствовать доставке генетического материала в митохондрии. Кроме того, одна соматическая клетка может содержать сотни митохондрий с большим количеством копий генома, что делает сомнительной возможность прямой митохондриальной доставки нуклеиновых кислот.Чтобы обойти эту трудность, группа доктора Гая в Майами в 2002 г. предложила технологию аллотопической экспрессии. В этой технологии используется трансген ядерной ДНК (яДНК), кодирующий митохондриальный белок MT-ND4 дикого типа. С помощью вектора аденоассоциированного вируса (AAV) трансген вводят в ядро ганглиозных клеток сетчатки. С помощью митохондриальной сигнальной последовательности информационная РНК затем перемещается из ядра на внешнюю мембрану митохондрий, где белок MT-ND4 дикого типа транслируется и включается в комплекс митохондриальной дыхательной цепи [Guy J, Qi X, Pallotti F, et al. Rescue of a mitochondrial deficiency causing Leber Hereditary Optic Neuropathy. Ann Neurol. 2002 Nov;52(5):534-42. doi: 10.1002/ana. 10354; Chi SC, Cheng HC, Wang AG. Leber Hereditary Optic Neuropathy: Molecular Pathophysiology and Updates on Gene Therapy. Biomedicines. 2022 Aug9;10(8): 1930. doi: 10.3390/biomedicines10081930; Artika I.M. Allotopic expression of mitochondrial genes: Basic strategy and progress // Genes Dis. Elsevier, 2020. Vol. 7, №4. P. 578-584].
В 2002 году Гай и соавторы использовали аденоассоциированный вирусный вектор (AAV), содержащий кодонно оптимизированную последовательность, кодирующую белок MT-ND4 и последовательность, кодирующую пептид митохондриальной локализации (MTS). MTS представлял собой N-концевую область либо (1) изоформы Р1 субъединицы с АТФ-синтазы человека (АТФс), содержащей всю 61-аминокислотную последовательность MTS вместе с пятью аминокислотами зрелого полипептида Р124, либо (2) Aldh, содержащий первый MTS из 19 аминокислот.Трансдуцированные описанными вирусами цибриды с мутацией гена MT-ND4 продуцировали в три раза больше АТФ, чем трансдуцированные пустыми вирусными частицами цибриды, что авторы интерпретировали как успешное восстановление функций комплекса I [Guy J, Qi X, Pallotti F, et al. Rescue of a mitochondrial deficiency causing Leber Hereditary Optic Neuropathy. Ann Neurol. 2002 Nov;52(5):534-42. doi: 10.1002/ana. 10354].
В 2016 году Фейер и соавторы провели первые клинические испытания (КИ) самокомплементарного AAV scAA-V2(Y444,500,730F)-P1ND4v2 на пяти пациентах с мутацией в гене MT-ND4, которым проводилась генная терапия на одном глазу. scAA-V2(Y444,500,730F)-P1ND4v2 содержит оптимизированную для транскрипции в ядре последовательность гена MT-ND4 и последовательность, кодирующую пептид митохондриальной локализации изоформы Р1 субъединицы с АТФ-синтазы. Ни у одного пациента не было зарегистрировано серьезных побочных явлений в ответ на терапию. У двух пациентов наблюдалось достоверное повышение остроты зрения. С анатомической точки зрения не было выявлено существенных различий в толщине слоев сетчатки [Feuer W.J. et al. Gene Therapy for Leber Hereditary Optic Neuropathy: Initial Results // Ophthalmology. Elsevier, 2016. Vol. 123, №3. P. 558-570].
В 2017 году компания GenSight Biologics проводила рандомизированное, двойное слепое, плацебо-контролируемое и многоцентровое клиническое исследование (КИ) III фазы. Пациентам в один глаз вводили GS010: рекомбинантный аденоассоциированный вирус, содержащий модифицированную кДНК, кодирующую человеческий митохондриальный белок MT-ND4 дикого типа и MTS СОХ10. Другой глаз подвергали фиктивной инъекции. В среднем у пациентов наблюдали улучшение остроты зрения. Аналогичное улучшение было зарегистрировано в другом, фиктивно инъецированном глазу. Это вызвало подозрение на перенос ДНК вирусного вектора из инъецированного глаза в другой глаз [Yu-Wai-Man P. et al. Bilateral visual improvement with unilateral gene therapy injection for Leber hereditary optic neuropathy // Sci. Transl. Med. American Association for the Advancement of Science, 2020. Vol.12, №573]. В июле 2019 III фаза КИ GS010 была успешно завершена.
В январе 2022 года Китайская компания Neurophth Biotechnology Limited получила одобрение Комитета Европейского агентства по лекарственным средствам (European Medicines Agency, ЕМА) на препарат NR082 (rAAV2-ND4) - новый рекомбинантный аденоассоциированный вирусный вектор серотипа 2, содержащий митохондриальный кодонно оптимизированный ген MT-ND4 под контролем промотора и энхансера цитомегаловируса.
Также в 2014 г. в Глазном институте им. Баскома Палмера Университета Майами было начато клиническое исследование фазы 1 scAAV2-P1ND4v2 для лечения ассоциированной с ND4 LHON. В предварительных результатах сообщалось о признаках эффективности у 2 пациентов из хронической группы и 4 пациентов из «острой» группы. В исследуемых когортах дозозависимого эффекта не наблюдали [Sahel J.A. et al. Gene Therapies for the Treatment of Leber Hereditary Optic Neuropathy // Int. Ophthalmol. Clin. Wolters Kluwer Health, 2021. Vol.61, №4. P. 195].
В заявке WO2019241206A1 раскрывается применение рекомбинантного вектора, кодирующего полипептид НАДН-дегидрогеназы 4 (ND4) человека и содержащего последовательность нуклеиновой кислоты, кодирующую MTS Сох10, полипептид NADH-дегидрогеназы 4 (ND4) и 3'UTR Сох10 при лечении наследственной оптической нейропатии Лебера (LHON).
В патенте CN109207520 В раскрыта рамка считывания для внутриядерной экспрессии митохондриального гена MT-ND4, которая включает ядерный промотор САМ, слитую последовательность, кодирующую пептид митохондриальной локализации СОХ10 и экспрессирующийся в ядре ген ND4, а также последовательность, заякоривающую мРНК на поверхности митохондрий.
Патент CN104450747 (Wuhan Neurophth Biological Technology Co., Ltd.), раскрывает ген ND4 для лечения LHON, в сочетании с последовательностями промотора CAG, Cox10 MTS и UTR, длина которой составляет 625 п. о., общая длина последовательности составляет 3824 п.н. AAV, несущий эту последовательность, вводят интравитреально для лечения LHON. Патент CN111073899 В того же правообладателя раскрывает слитую нуклеиновую кислоту, которая кодирует от 5'-конца до 3'-конца: MTS (СОХ10), ND4, UTR.
Патент CN110876269 В, раскрывает различные варианты последовательности нуклеиновой кислоты, кодирующей белок ND4, а также различные варианты нуклеотидной последовательности, кодирующей MTS, такие как нативный и оптимизированный MTS СОХ10, нативный и оптимизированный MTS ОРА1. Оптимизированные последовательности были разработаны для повышения эффективности транскрипции и трансляции. Рекомбинантная нуклеиновая кислота содержала последовательности, кодирующие MTS и ND4, а также нетранслируемую область 3'UTR. Синтетическую рекомбинантную нуклеиновую кислоту встраивали в вектор AAV2 для дальнейшего использования при введении в клетки-мишени. Другая оптимизированная последовательность ND4, слитая с последовательностью, кодирующей MTS СОХ10 или ОРА1 и/или 3'UTR раскрыта в патенте CN110699367 В CN109970861B.
Патент ЕР1880008 В1 (Institut National de la Sante et de la Recherche Medicale INSERM) раскрывает экспрессионный вектор, адаптированный для стабильной и эффективной доставки белка в митохондрии. Вектор содержит последовательность, кодирующую MTS, в частности СОХ10 (цитохром-с-оксидазы 10) или SOD2 (супероксиддисмутазы 2), кодирующую последовательность белка, в частности ND4, и последовательность с 3'-конца этих последовательностей, в частности SV40 3'-UTR или СОХ10 3'-UTR. Указанный вектор использует посттрансляционный путь импорта, однако не использует иные последовательности пептидов митохондриальной локализации кроме как СОХ10 и SOD2.
Однако существующие аналоги недостаточно эффективны. Так, например, компания Gensight Biologics SA 20 апреля 2023 года отозвала заявку на регистрацию препарата Lumevoq для лечения потери зрения, вызванной заболеванием глаз, известным как наследственная оптическая нейропатия Лебера. Результаты исследований не показали существенной разницы в зрении в глазах, которым вводили Lumevoq, и в глазах, которым вводили имитацию или плацебо. Кроме того, исследования не предоставили достаточных доказательств того, что введение Lumevoq в оба глаза принесет пользу пациентам [https://www.ema.europa.eu/en/medicines/human/withdrawn-applications/lumevoq, дата обращения 09.08.2023].
Технической задачей, на решение которой направлено настоящее изобретение, является решение как минимум одной из вышеуказанных в уровне техники проблем.
Сущность изобретения
Технической решением является использование описанного признаками в пунктах формулы изобретения.
Одной из возможных технических задач, на решение которой может быть направлено настоящее изобретение, являлось расширение арсенала технических средств, направленных на снижение последствий нарушения функции митохондрий в клетках с мутацией в гене MT-ND4. Изобретение может быть использовано при генной терапии наследственной оптической нейропатии Лебера.
Техническим результатом, достигаемым при осуществлении настоящего изобретения, является снижение последствий нарушения функции митохондрий в клетках с мутацией в гене MT-ND4.
Задача может решаться путем создания последовательности нуклеотидов для аллотопической экспрессии гена MT-ND4 в клетках. Технический результат может достигаться созданием кодонно оптимизированной последовательности ND4opt (Seq Id No: 1), кодирующей нормальный белок MT-ND4.
В одном из вариантов осуществления настоящего изобретения предлагается нуклеиновая кислота для аллотопической экспрессии гена MT-ND4, содержащая последовательность нуклеотидов Seq Id No.: 1 и последовательность, кодирующую пептид митохондриальной локализации субъединицы 8 цитохром-с-оксидазы (белка Сох8).
В одном из вариантов осуществления настоящего изобретения пептид митохондриальной локализации белка Сох8 содержит последовательность Seq Id No.:6. В еще одном варианте осуществления изобретения последовательность Seq Id No.:5 кодирует пептид митохондриальной локализации белка Сох8.
В другом варианте осуществления изобретения предлагается экспрессионный вектор для экспрессии в эукариотических клетках, содержащий нуклеиновую кислоту для аллотопической экспрессии гена MT-ND4 по настоящему изобретению.
В другом варианте осуществления изобретения предлагается плазмидный экспрессионный вектор, содержащий в соответствии с физической и генетической картой, представленной на Фиг. 4, следующие элементы:
- участок начала репликации ori;
- левый инвертированный концевой повтор ITR;
- цитомегаловирусный промотор CMV promoter;
- последовательность интрона гена b-глобина человека;
- последовательность, кодирующую последовательность SEQ Id No.:6 пептида митохондриальной локализации (MTS) белка СОХ8;
- последовательность ND4opt Seq Id No.: 1;
- последовательность сигнала полиаденилирования poly(A);
- правый инвертированный концевой повтор ITR;
- участок начала репликации для упаковки в фаговые частицы f1 ori;
- промотор гена устойчивости к ампициллину AmpR promoter;
- ген устойчивости к антибиотику ампициллину AmpR.
В еще одном из вариантов осуществления изобретения упомянутый вектор содержит последовательность SEQ Id No.:5 в качестве последовательности, кодирующей пептид митохондриальной локализации (MTS) гена СОХ8 (SEQ Id No.:6).
В другом варианте осуществления изобретения упомянутый вектор представляет собой вирусный экспрессионный вектор, в частном случае аденоассоциированный вирус, в частном случае аденоассоциированный вирус 6 серотипа.
В другом варианте осуществления изобретения в качестве вирусного экспрессионного вектора предлагается аденоассоциированный вирус 6 серотипа, нарабатываемый в суспензионных клеточных культурах HEK293.
В другом варианте осуществления изобретения предлагается применение экспрессионного вектора по настоящему изобретению для лечения наследственной оптической нейропатии Лебера.
Введение в клетки с мутацией в гене MT-ND4 нуклеиновой кислоты по настоящему изобретению приводит к статистически значимому снижению уровня таких маркеров окислительного стресса как содержание в клетках активных форм кислорода (АФК) и ионов кальция в митохондриях. Это свидетельствует о снижении последствий нарушения функции митохондрий. Снижение уровня маркеров окислительного стресса также косвенно подтверждает восстановление функции НАДН-дегидрогеназного комплекса в клетках с мутацией в гене MT-ND4. Таким образом, технический результат: снижение последствий нарушения функции митохондрий в клетках с мутацией в гене MT-ND4, - может быть достигнут при осуществлении настоящего изобретения.
Изобретение иллюстрируются следующими графическими материалами: На Фиг. 1 представлена структурная карта рекомбинантного плазмидного экспрессионного вектора pAAV-ND4opt, экспрессирующего оптимизированную последовательность ND4opt. ДНК с нуклеотидной последовательностью ND4opt была синтезирована и встроена в плазмиду pAAV-MCS по сайтам рестрикции (BamHI и HindIII) под контролем CMV промотора.
На Фиг. 2 представлены пептиды митохондриальной локализации различных белков.
На Фиг. 3 представлена структурная карта рекомбинантного плазмидного экспрессионного вектора pAAV-MTS4-ND4opt, экспрессирующего кодонно оптимизированный вариант гена MT-ND4 (ND4opt) с последовательностью, кодирующей MTS СОХ4. ДНК с последовательностью, кодирующей MTS СОХ4, слитой с ND4opt, была синтезирована и встроена в плазмиду pAAV-MCS по сайтам рестрикции (BamHI и HindIII) под контролем CMV промотора.
На Фиг. 4 представлена структурная карта рекомбинантного плазмидного экспрессионного вектора pAAV-MTS8-ND4opt, экспрессирующего кодонно оптимизированный вариант гена MT-ND4 (ND4opt) с последовательностью, кодирующей MTS СОХ8. ДНК с последовательностью, кодирующей MTS СОХ8, слитой с ND4opt была синтезирована и встроена в плазмиду pAAV-MCS по сайтам рестрикции (BamHI и HindIII) под контролем CMV промотора.
На Фиг. 5 представлена структурная карта рекомбинантного плазмидного экспрессионного вектора pAAV-MTS8(N)-ND4opt, экспрессирующего кодонно оптимизированный вариант гена MT-ND4 (ND4opt) с последовательностью, кодирующей MTS COX8(N). ДНК с последовательностью, кодирующей MTS COX8(N), слитой с ND4opt, была синтезирована и встроена в плазмиду pAAV-MCS по сайтам рестрикции (BamHI и HindIII) под контролем CMV промотора.
На Фиг. 6 представлена структурная карта рекомбинантного плазмидного экспрессионного вектора pAAV-MTS10-ND4opt, экспрессирующего кодонно оптимизированный вариант гена MT-ND4 (ND4opt) с последовательностью, кодирующей MTS СОХ10. ДНК с последовательностью, кодирующей MTS СОХ10, слитой c ND4opt, была синтезирована и встроена в плазмиду pAAV-MCS по сайтам рестрикции (BamHI и HindIII) под контролем CMV промотора.
На Фиг. 7 представлены результаты оценки флуоресценции после трансфекции клеток HEK293 плазмидными экспрессионными векторами: pHyper-dMito и вектором, экспрессирующим последовательность, кодирующую вариант MTS с ND4opt. По оси ординат - количество флуоресцирующих клеток.
pHyper-dMito - трансфекция вектором pHyper-dMito;
cox8k-ND4 - трансфекция векторами pHyper-dMito и pAAV-MTS8-ND4opt;
cox8n-ND4 - трансфекция векторами pHyper-dMito и pAAV-MTS8(N)-ND4opt;
cox10-ND4 - трансфекция векторами pHyper-dMito и pAAV-MTS10-ND4opt;
cox4-ND4 - трансфекция векторами pHyper-dMito и pAAV-MTS4-ND4opt;
pAAV-MCS - трансфекция исходным вектором pAAV-MCS, не экспрессирующим ND4opt.
На Фиг. 8 представлены результаты оценки флуоресценции при трансфекции фибробластов клеточной модели LHON с мутацией в гене ND4 плазмидными экспрессионными векторами: pHyper-dMito и вектором, экспрессирующим последовательность, кодирующую вариант MTS с ND4opt. По оси ординат - количество флуоресцирующих клеток.
pHyper-dMito - трансфекция вектором pHyper-dMito;
cox8k-ND4 - трансфекция векторами pHyper-dMito и pAAV-MTS8-ND4opt;
cox8n-ND4 - трансфекция векторами pHyper-dMito и pAAV-MTS8(N)-ND4opt;
cox10-ND4 - трансфекция векторами pHyper-dMito и pAAV-MTS10-ND4opt;
cox4-ND4 - трансфекция векторами pHyper-dMito и pAAV-MTS4-ND4opt;
pAAV-MCS -трансфекция исходным вектором pAAV-MCS, не экспрессирующим ND4opt.
На Фиг. 9 представлены результаты оценки флуоресценции при трансфекции фибробластов клеточной модели LHON с мутацией в гене ND4 двумя плазмидными экспрессионными векторами: pCase12-Mito и вектором, экспрессирующим последовательность, кодирующую вариант MTS с ND4opt. По оси ординат количество флуоресцирующих клеток.
pCase12-Mito - трансфекция вектором pCase12-Mito;
cox8k-ND4 трансфекция векторами pCase12-Mito и pAAV-MTS8-ND4opt;
cox8n-ND4 трансфекция векторами pCase12-Mito и pAAV-MTS8(N)-ND4opt;
cox10-ND4 - трансфекция векторами pCase12-Mito и pAAV-MTS10-ND4opt;
cox4-ND4 - трансфекция векторами pCase12-Mito и pAAV-MTS4-ND4opt;
pAAV-MCS - трансфекция исходным вектором pAAV-MCS, не экспрессирующим ND4opt.
На Фиг. 10 представлены результаты оценки флуоресценции DCF при трансфекции фибробластов клеточной модели LHON с мутацией в гене ND4 плазмидными экспрессионными векторами, экспрессирующими последовательность, кодирующую вариант MTS с ND4opt. По оси ординат - уровень флюоресценции в условных единицах после нормирования на 100 тысяч клеток.
Клетки (К) - клетки без трансфекции;
cox8k-ND4 трансфекция вектором pAAV-MTS8-ND4opt;
cox8n-ND4 -трансфекция вектором pAAV-MTS8(N)-ND4opt;
DNAJC30-ND4 -трансфекция вектором pAAV-MTS-DNAJC30-ND4opt;
cox10-ND4 трансфекция вектором pAAV-MTS 10-ND4opt;
cox4-ND4 -трансфекция вектором pAAV-MTS4-ND4opt;
pAAV - трансфекция исходным вектором pAAV-MCS, не экспрессирующим ND4opt.
На Фиг. 11 представлены результаты оценки флуоресценции DCF при трансдукции фибробластов клеточной модели LHON аденоассоциированным вирусным вектором, экспрессирующим последовательность, кодирующую вариант MTS с ND4opt. По оси ординат уровень флюоресценции в условных единицах после нормирования на 100 тысяч клеток.
Клетки (К) - клетки без трансдукции;
AAV8k трансдукция вектором rAAV-MTS8-ND4;
AAV10 - трансдукция вектором rAAV-MTS10-ND4.
Детальное описание изобретения
Если не указано иначе, предполагается, что все термины, обозначения и другие научные термины, используемые в данной заявке, имеют значения, которые обычно понимают специалисты в области, к которой относится настоящее изобретение. В некоторых случаях определения терминов с общепринятыми значениями приведены в данной заявке для ясности и/или для быстрой справки и понимания, и включение таких определений в настоящее описание не должно истолковываться как наличие существенного отличия значения термина от обычно подразумеваемого в данной области.
Кроме того, если по контексту не требуется иное, термины в единственном числе включают в себя термины во множественном числе, и термины во множественном числе включают в себя термины в единственном числе. Как правило, используемая классификация и методы культивирования клеток, молекулярной биологии, иммунологии, микробиологии, генетики, аналитической химии, химии органического синтеза, медицинской и фармацевтической химии, а также гибридизации и химии белка и нуклеиновых кислот, описанные в настоящем документе, хорошо известны специалистам и широко применяются в данной области. Ферментативные реакции и способы очистки осуществляют в соответствии с инструкциями производителя, как это обычно осуществляется в данной области, или как описано в настоящем документе.
В настоящем изобретении предлагается нуклеиновая кислота для аллотопической экспрессии гена MT-ND4, содержащая последовательность нуклеотидов Seq Id No.: 1 и последовательность, кодирующую пептид митохондриальной локализации субъединицы 8 цитохром-с-оксидазы (белка Сох8).
Последовательность нуклеотидов Seq Id No.: 1 кодирует нормальный белок МТ-ND4 человека и кодонно оптимизирована на основе последовательности гена MT-ND4 дикого типа. На основе последовательности гена MT-ND4 дикого типа (NC 012920.1, Gene ID: 4538) была проведена оптимизация кодонов для того, чтобы обеспечить аллотопическую экспрессию митохондриального гена MT-ND4. Следовательно, кодонно оптимизированная последовательность гена MT-ND4 по настоящему изобретению, также называемая в описании изобретения ND4opt, кодирует нормальный белок МТ-ND4, который идентичен белку MT-ND4, синтезируемому в митохондриях. Кодонно оптимизированная последовательность ND4opt на 73% гомологична митохондриальной версии гена ND4, тогда как аминокислотные последовательности, кодируемые обеими нуклеотидными последовательностями, идентичны.
Белок Сох8 представляет собой субъединицу 8 цитохром-с-оксидазы, его аминокислотная последовательность содержит участок, отвечающий за митохондриальную локализацию - пептид митохондриальной локализации (MTS).
В одном из вариантов осуществления настоящего изобретения пептид митохондриальной локализации белка Сох8 содержит последовательность Seq Id No.:6. В еще одном варианте осуществления изобретения последовательность Seq Id No.:5 кодирует пептид митохондриальной локализации белка Сох8.
Термин «кодонно оптимизированный» обозначает последовательность нуклеотидов, в которой была произведена замена одного или более кодонов на синонимичные без изменения последовательности белка, синтезированного с матрицы этой последовательности.
Оптимизация кодонов нами выполнена с учетом представленности кодонов и тРНК в клетках человека с уровнем относительной адаптивности не менее 50%. Разработанная кодонно оптимизированная последовательность лишь на 73% идентична последовательности MT-ND4 человека. Оптимизация кодонов при создании настоящего изобретения была выполнена с учетом вторичной структуры РНК и стабильности шпилек молекулы РНК, для исключения стоп-кодонов и несинонимичных кодонов, а также при проведении оптимизации учитывали структурные особенности белка ND4, в частности был проведен анализ неструктурированных регионов белка с целью выбора оптимальных регионов для оптимизации с помощью IUPred2 [https://iupred2a.elte.hu, дата обращения 16.06.2023].
В результате оптимизации кодонов было получено несколько вариантов последовательности нуклеотидов, кодирующих ND4, из которых была выбрана последовательность нуклеотидов Seq Id No.: 1 с оптимальными свойствами, предсказанными in silico. ДНК с последовательностью нуклеотидов по настоящему изобретению успешно синтезирована и введена в плазмидный экспрессионный вектор на основе pAAV и вирусный экспрессионный вектор на основе аденоассоциированного вируса. ДНК с последовательностью нуклеотидов по настоящему изобретению стабильно экспрессируется как в клетках адгезионной культуры, так и в клетках суспензионной культуры.
Термин «оптимизация кодонов» означает экспериментальный подход для улучшения кодонного состава рекомбинантного гена на основе различных критериев без изменения аминокислотной последовательности. Оптимизация кодонов возможна благодаря вырожденному генетическому коду, означающему, что большинство аминокислот кодируется более чем одним кодоном. Большинство подходов к оптимизации кодонов заключается в избегании использования редких кодонов. Также направлением оптимизации кодонов может быть выявление элементов нестабильности мРНК, вторичных структур мРНК, повторов последовательностей, внутренних сайтов входа в рибосому, промоторных последовательностей, предполагаемых сайтов сплайсинга и пр. примеры технологий такой оптимизации писаны в статье [Gao, W. et al. (2004) UpGene: Application of a web-based DNA codon optimization algorithm. Biotechnol. Prog. 20, 443 448; Raab, D. et al. (2010) The GeneOptimizer Algorithm: using a sliding window approach to cope with the vast sequence space in multiparameter DNA sequence optimization. Syst. Synth. Biol. 4, 215-225; Gaspar, P. et al. (2012) EuGene: maximizing synthetic gene design for heterologous expression. Bioinformatics 28, 2683-2684; Fath, S. et al. (2011) Multiparameter RNA and codon optimization: a standardized tool to assess and enhance autologous mammalian gene expression. PLoS ONE 6, e17596], текст которой инкорпорирован в настоящее описание посредством ссылки. Частоты встречаемости синонимических кодонов различаются у разных организмов и даже в разных клетках одного и того же организма. Они декодируются рибосомами с разной скоростью, так как соответствующие им тРНК также присутствуют в разных клетках в разных количествах, как показано в статье [Dana A., Tuller Т. (2014) The effect of tRNA levels on decoding times of mRNA codons. Nucl. Acids Res. 42, 9171-9181], текст которой инкорпорирован в настоящее описание посредством ссылки. Трансляция в митохондриях отклоняется от универсального генетического кода, используя механизмы и частоты кодонов, более сходные с их α-протеобактериальными предками, чем с ядерным геномом млекопитающих, поэтому для успешной экспрессии в цитозоле митохондриальных генов используется оптимизация кодонов для перекодирования последовательности митохондриального гена в универсальный код [Lewis CJ, Dixit В, Batiuk Е, et al. Codon optimization is an essential parameter for the efficient allotopic expression of mtDNA genes. Redox Biol. 2020 Feb;30:101429. doi: 10.1016/j.redox.2020.101429].
Термин «аллотопическая экспрессия» описывает способ экспрессии митохондриальных генов, при котором последовательность митохондриального гена кодонно оптимизируют в соответствии с универсальным кодом для того, чтобы транскрипция проходила в ядре с использованием эукариотического транскрипционного аппарата. При этом к последовательности митохондриального гена интереса, как правило, присоединяют последовательность, кодирующую гетерологичный пептид митохондриальной локализации (MTS) для того, чтобы полученный в результате аллотопической экспрессии белок локализовался в митохондрии. В последовательностях ядерных генов, кодирующих митохондриальные белки, также закодированы и последовательности MTS.
Нуклеиновая кислота по настоящему изобретению, в результате экспрессии в клетках с мутацией гена MT-ND4, приводит к локализации нормального белка MT-ND4 в митохондриях, благодаря наличию последовательности пептида митохондриальной локализации белка СОХ8, и восполняет подавленную функцию аутологичного НАДН-дегидрогеназного комплекса. В результате снижаются последствия нарушения функции митохондрий в клетках с мутацией в гене MT-ND4, что выражается в снижении уровня маркеров окислительного стресса. При этом нуклеиновая кислота по настоящему изобретению может быть доставлена в клетки любым известным из уровня техники способом доставки и экспрессии, в том числе путем прямого введения, а также путем доставки в клетку с помощью известных из уровня техники экспрессионных векторов. Выбор способа доставки нуклеиновой кислоты по настоящему изобретению не влияет на достижение технического результата.
Введение в клетки с мутацией в гене MT-ND4 нуклеиновой кислоты для аллотопической экспрессии нормального белка MT-ND4 по настоящему изобретению снижало количество АФК в клетках на 55%. Это говорит о том, что нуклеиновая кислота по настоящему изобретению экспрессируется в клетках с мутацией в гене MT-ND4, приводя к накоплению нормального белка MT-ND4 и снижая последствия недостаточной работы НАДН-дегидрогеназного комплекса. Этот эффект не ограничивается клетками с конкретной мутацией и может распространяться на клетки с любой мутацией в гене MT-ND4, поскольку замещает мутантный белок функциональным (здоровым). Нуклеиновая кислота по настоящему изобретению таким образом может быть использована для заместительной генной терапии LHON.
Нуклеиновая кислота с заявленной последовательностью может быть получена любым известным из уровня техники способом, включая, в качестве неограничивающих примеров, рекомбинантные способы, такие как клонирование последовательностей нуклеиновой кислоты из рекомбинантной библиотеки или генома клетки, использование обычной технологии клонирования и ПЦР и другими, а также способами химического синтеза.
Нуклеиновая кислота по настоящему изобретению может быть введена в плазмидный экспрессионный вектор, например, на основе pAAV. Введение плазмидного экспрессионного вектора, содержащего последовательность нуклеотидов по настоящему изобретению, в клетки НЕК293Т путем трансфекции приводит к экспрессии гетерологичного гена MT-ND4 (ND4opt), при этом уровень активных форм кислорода (АФК) в митохондриях трансфицированных клеток снижается по меньшей мере в 2 раза.
Термин «вектор» обозначает молекулу нуклеиновой кислоты, используемую для передачи генетического материала внутрь клетки. Термин «экспрессионный вектор» обозначает вектор, содержащий промоторную и другие регуляторные последовательности, обеспечивающие эффективную транскрипцию рекомбинантного гена с последующей трансляцией мРНК и образованием рекомбинантного белка. Используемые плазмидные и экспрессионные векторы, приведенные ниже, используются для примера и не ограничивают объем прав настоящего изобретения. Термин «экспрессия гена» обозначает преобразование наследственной информации, зашифрованной в последовательности нуклеотидов гена, в функциональный продукт - РНК или белок.
В одном из вариантов осуществления изобретения предлагается экспрессионный вектор для экспрессии в эукариотических клетках, содержащий нуклеиновую кислоту по настоящему изобретению. Нуклеиновая кислота по настоящему изобретению может быть введена в любой известный из уровня техники вектор, в том числе в плазмидный, вирусный, в том числе на основе вируса SV40, аденовирусов, герпесвирусов, ретровирусов, лентивирусов, аденоассоциированного и других вирусов. Векторы, в которые может быть введена нуклеиновая кислота по настоящему изобретению, не ограничивается этим списком. Выбор экспрессионного вектора определяется задачами, для которых вектор будет в дальнейшем использован и не ограничивает объем настоящего изобретения, способы получения векторов известны из уровня техники, векторы, содержащие нуклеиновую кислоту по настоящему изобретению могут быть получены специалистами в области генетической инженерии.
В варианте осуществления изобретения предлагается плазмидный экспрессионный вектор, содержащий элементы в соответствии с физической и генетической картой, представленной на Фиг. 4. Термин «промотор», используемый в настоящем документе, в частности, относится к последовательности нуклеотидов ДНК, узнаваемой РНК-полимеразой, на которой происходит инициация транскрипции элемента, с которым функционально связан промотор. Промотор может также сопровождаться энхансером.
Энхансеры усиливают активность промотора и стимулируют процесс транскрипции. Для получения большого количества белка в эукариотических клетках и, в частности, в клетках человека, целесообразно использовать сильные промоторы, активные в клетках-мишенях. Сильные конститутивные промоторы, способные инициировать экспрессию рекомбинантного гена в разных типах клеток, хорошо известны в данной области. В одном из вариантов осуществления изобретения в качестве промотора используют промотор цитомегаловируса (CMV promoter).
В варианте осуществления изобретения плазмидный экспрессионный вектор включает следующие элементы в направлении от 5'-конца к 3'-концу:
- участок начала репликации ori;
- левый инвертированный концевой повтор ITR;
- цитомегаловирусный промотор CMV promoter;
- последовательность интрона гена b-глобина человека;
- последовательность, кодирующую последовательность SEQ Id No.:6 пептида митохондриальной локализации (MTS) белка СОХ8;
- последовательность ND4opt Seq Id No.: 1;
- последовательность сигнала полиаденилирования poly(A);
- правый инвертированный концевой повтор ITR;
- промотор гена устойчивости к ампициллину AmpR promoter;
- ген устойчивости к антибиотику ампициллину AmpR.
В еще одном из вариантов осуществления изобретения упомянутый вектор содержит последовательность SEQ Id No.:5 в качестве последовательности, кодирующей пептид митохондриальной локализации (MTS) белка СОХ8 (SEQ Id No.:6).
Трансфекция клеток плазмидными экспрессионными векторами по настоящему изобретению, приводит к стабильной экспрессии ND4opt и снижению уровня АФК и митохондриального уровня кальция по меньшей мере в 2 раза.
В одном из вариантов осуществления изобретения предлагается вирусный экспрессионный вектор, содержащий нуклеиновую кислоту по настоящему изобретению. Вектор может быть вектором аденоассоциированного вируса (AAV), аденовирусным вектором, ретровирусом или лентивирусным вектором на основе вируса иммунодефицита человека и других вирусов. AAV и лентивирусы могут обеспечивать длительную экспрессию, в то время как аденовирус может обеспечивать временную экспрессию.
В одном из вариантов осуществления изобретения предлагается аденоассоциированный вирус, содержащий нуклеиновую кислоту по настоящему изобретению, в качестве вирусного экспрессионного вектора. Примеры таких векторов на основе AAV можно найти в статье: Peters, С.W., Maguire, С.А., & Hanlon, K.S. (2021). Delivering AAV to the Central Nervous and Sensory Systems. Trends in Pharmacological Sciences, 42(6), 461 474. Векторы на AAV признаются эффективными для генной терапии производных нервной ткани, в том числе тканей сенсорных органов, таких как глаз. AAV представляет собой небольшой, неспособный к самостоятельной репликации, вирус, не имеющий оболочки, размером около 20 нм. У человека и приматов описано множество различных серотипов AAV. Геном всех известных серотипов AAV организован сходно: он представляет собой линейную одноцепочечную молекулу ДНК размером менее чем примерно 5000 нуклеотидов (нт). Инвертированные концевые повторы (англ. inverted terminal repeats, ITR) ограничивают внутри себя уникальные нуклеотидные последовательности, кодирующие капсидные белки (Сар) и белки репликации (Rep). Ген Сар кодирует белки VP (VP1, VP2 и VP3), которые образуют капсид. При образовании рекомбинантного вектора AAV кассету экспрессии, не содержащую гены Rep и ограниченную ITR, помещают в капсид AAV. Рекомбинантный AAV не способен к репликации и в настоящее время признается одним из самых безопасных и широко используемых вирусных экспрессионных векторов для переноса генов in vivo. Векторы на основе AAV могут проникать в клетки различных тканей, обеспечивая стабильную экспрессию гетерологичного гена. Эти вирусы непатогенны и обладают низкой иммуногенностью [High KA, Aubourg P. rAAV human trial experience. Methods Mol Biol. 2011;807:429-57]. Выбор серотипа AAV определяется целями и задачами исследования, типом клеток, для трансдукции которых разрабатывается вирус и не ограничивает объема прав настоящего изобретения.
В одном из вариантов осуществления изобретения предлагается аденоассоциированный вирус 6 серотипа, содержащий нуклеиновую кислоту по настоящему изобретению, в качестве вирусного экспрессионного вектора. Все известные серотипы аденоассоциированных вирусов могут инфицировать клетки многих видов тканей. Тканевая специфичность определяется серотипом белков капсида, поэтому векторы на основе аденоассоциированого вируса конструируют, задавая необходимый серотип. Использование вирусного экспрессионного вектора на основе AAV 6 серотипа, содержащего нуклеиновую кислоту по настоящему изобретению, приводит к стабильной экспрессии ND4opt в трансдуцированных ими клетках, снижая при этом уровень АФК на ~25%.
В одном из вариантов осуществления изобретения в качестве вирусного экспрессионного вектора предлагается аденоассоциированный вирус 6 серотипа, содержащий нуклеиновую кислоту по настоящему изобретению, нарабатываемый в суспензионных клеточных культурах HEK293. Введение плазмидного экспрессионного вектора, содержащего нуклеиновую кислоту по настоящему изобретению, совместно с вектором pHelper и вектором pRC2/6 в клетки суспензионной клеточной культуры HEK293 приводит к продукции вирусных экспрессионных векторов, представляющих собой аденоассоциированный вирус 6 серотипа. Было показано, что при трансдукции клеток полученными AAV-векторами, содержащими нуклеиновую кислоту по настоящему изобретению, уровень АФК в клетках снижается на ~25%.
В одном из вариантов осуществления изобретения предлагается применение экспрессионного вектора по настоящему изобретению, для лечения наследственной оптической нейропатии Лебера.
Для моделирования LHON in vitro и подбора лечения можно использовать методы, оценивающие метаболическое состояние клеток и факторы, которые являются следствием нарушения функций митохондрий, например накопление внутриклеточного кальция или активных форм кислорода, а в качестве способа коррекции заболевания - использовать доставку терапевтического гена в ядро с помощью хорошо зарекомендовавших себя в клинических исследованиях AAV-векторов и аллотопической экспрессии трансгена за счет использования пептида митохондриальной локализации. Однако не очевидно, будет ли аллотопическая экспрессия положительно влиять на восстановление метаболического состояния клеток и зависит ли это от типа пептида митохондриальной локализации.
Экспрессионные векторы по настоящему изобретению демонстрируют стабильную и высокую экспрессию ND4opt в клетках. Это свидетельствует в пользу того, что введение любого из заявленных экспрессионных векторов по настоящему изобретению в ткани глаза может привести к стабильной экспрессии ND4opt с последовательностью Seq Id No.:1 и восполнению недостатка белка MT-ND4 в клетках и тканях глаза, и, следовательно, восстановлению его функции и стабилизации зрения при наследственной оптической нейропатии Лебера. Введение плазмидного и вирусного векторов по настоящему изобретению в клетки с природной мутацией гена MT-ND4 приводят к накоплению нормального белка MT-ND4 и восстановлению функции митохондрий, выражающемуся в снижении уровня маркеров окислительного стресса.
Сущность и промышленная применимость изобретения поясняются следующими примерами:
Пример 1. Создание плазмидных экспрессионных векторов.
Пример 1.1. Оптимизация кодонов последовательности нуклеотидов гена.MT-ND4.
Аннотацию кодирующей последовательности гена MT-ND4 проводили с использованием сервиса NCBI/CCD. Для выравнивания нуклеотидных последовательностей применяли инструмент UniPro UGENE [Okonechnikov K, Golosova О, Fursov М; UGENE team. Unipro UGENE: a unified bioinformatics toolkit. Bioinformatics. 2012 Apr 15;28(8): 1166-7. doi: 10.1093/bioinformatics/bts091]. Трансляцию нуклеотидной последовательности в аминокислотную проводили при помощи инструмента EMBOSS Transeq [https://www.ebi.ac.uk/Tools/st/emboss_transeq/, дата обращения 08.08.2023]. Поскольку существуют различия в кодировании митохондриальных и ядерных генов, а клонируемый трансген экспрессируется в ядерной системе, была проведена оптимизация последовательности нуклеотидов для исключения стоп-кодонов и несинонимичных кодонов. Для изучения стабильности транскрипта была использована программа RNA fold [http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi, дата обращения 16.06.2023]. Нуклеиновую кислоту с оптимизированной последовательностью гена MT-ND4 (ND4opt, SEQ Id No.: 1), кодирующую последовательность белка MT-ND4 (SEQ Id No.: 2), синтезировали на заказ при помощи сервиса в компании TopGenetech (Канада). Дополнительно был проведен анализ неструктурированных регионов белка с целью выбора оптимальных регионов для оптимизации с помощью IUPred2 [https://iupred2a.elte.hu, дата обращения 16.06.2023].
Пример 1.2. Получение экспрессионного вектора для аллотопической экспрессии ND4opt
В качестве плазмидного экспрессионного вектора использовали pAAV-MCS (cat#VPK-410, Cell biolabs). В качестве матрицы для амплификации использовали ДНК с оптимизированной последовательностью гена MT-ND4 (ND4opt), для полимеразной цепной реакции использовали полимеразу Q5 (New England Biolabs Inc., США). Реакционную смесь для ПЦР готовили согласно рекомендациям производителя.
Проводили ПЦР со следующими параметрами:
предварительное плавление ДНК при 98°С в течение 1 минуты, 25 циклов амплификации, включающих плавление в течение 10 секунд при 98°С, отжиг в течение 15 секунд при 58°С и элонгацию при 72°С в течение 30 секунд, конечную элонгацию при 72°С в течение 3 минут.
Условия проведения ПЦР:
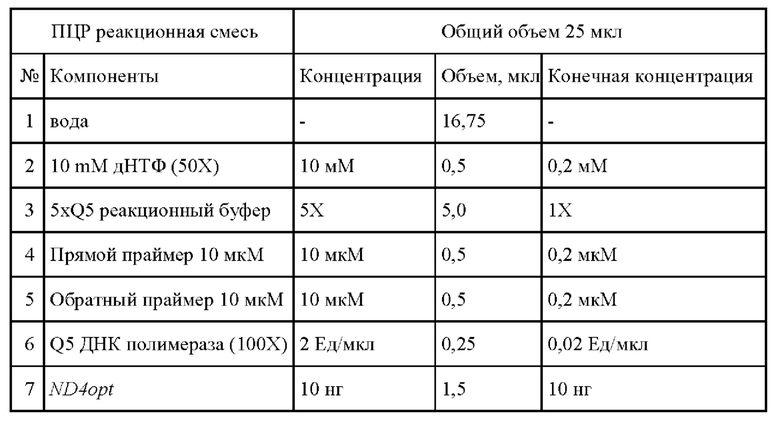
Встраивание последовательности ND4opt проводили по методике, рекомендуемой производителем вектора, с использованием эндонуклеаз рестрикции BamHI и HindIII (New England Biolabs Inc., США) и T4 ДНК-лигазы по стандартным протоколам.
В результате был получен рекомбинантный плазмидный экспрессионный вектор pAAV-ND4opt (Фиг. 1), содержащий в соответствии с физической и генетической картой, представленной на Фиг. 1, следующие элементы:
- участок начала репликации ori;
- левый инвертированный концевой повтор ITR;
- цитомегаловирусный промотор CMV promoter;
- последовательность интрона гена b-глобина человека;
- последовательность ND4opt Seq Id No.: 1;
- последовательность сигнала полиаденилирования poly(A);
- правый инвертированный концевой повтор ITR;
- промотор гена устойчивости к ампициллину AmpR promoter;
- ген устойчивости к антибиотику ампициллину AmpR.
Пример 1.3. Подбор и клонирование последовательностей для направленного транспорта MT-ND4 в митохондрии
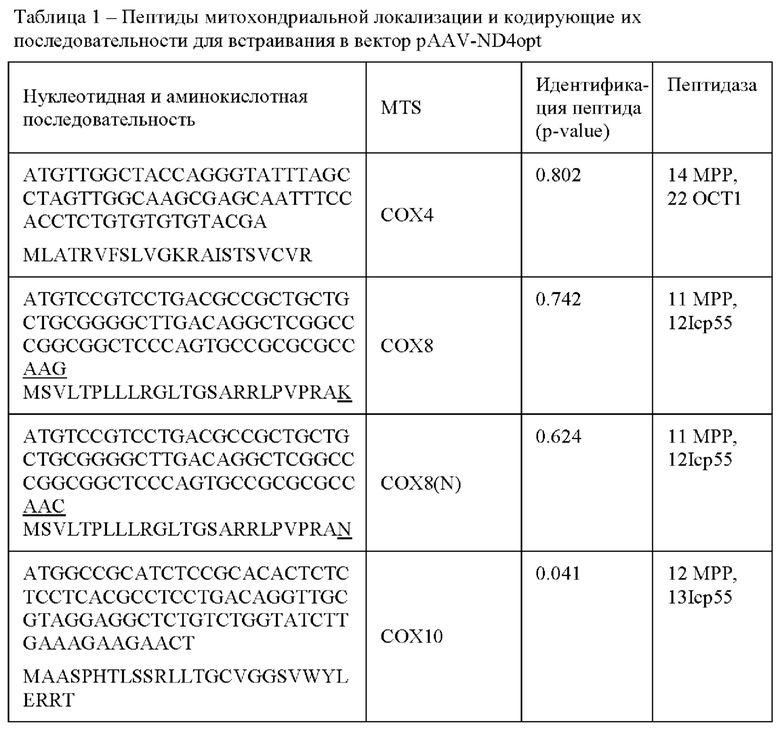
Пептиды митохондриальной локализации (MTS или MLS) цитохром С-оксидазы являются одними из наиболее хорошо изученных и часто используемых для доставки белков в митохондрии. MTS у цитохромоксидаз это обычно короткие пептидные последовательности (25-30 аминокислот) расположенные на N-участке полипептида. Идентифицировали MTS, а также проверяли мутационный ландшафт и эффективность процессинга MTS в MitaViewer [http://132.216.58.173/MTSvieweRl/, дата обращения 16.06.2023] и MitoFates [https://mitf.cbrc.pj.aist.go.jp/MitoFates/cgi-bin/top.cgi/, дата обращения 16.06.2023]. Результаты анализа суммированы в таблице 1 и Фиг. 2. Для СОХ8 были разработаны два варианта MTS: с лизином (К) в 25 позиции MTS и с заменой аминокислоты лизина (К) на аспарагин (N) в 25 позиции MTS. Позиция 25 в MTS СОХ8 является терминальной и критичной для дальнейшего процессинга белка.
Плазмидные экспрессионные векторы pAAV-MTS-ND4opt, экспрессирующие ND4opt с одним из вариантов последовательности, кодирующей MTS, были получены встраиванием последовательности MTS-ND4opt в последовательность плазмидного вектора pAAV для сборки вируса. ДНК с последовательностью MTS-ND4opt получали методом ПЦР с перекрыванием последовательностей (splicing by overlap extension / splicing by overhang extension (SOE) PCR, принцип метода описан, например, в работе: Horton RM, Но SN, Pullen JK, et al. Gene splicing by overlap extension. Methods Enzymol. 1993;217:270-9. doi: 10.1016/0076-6879(93)17067-f).
ДНК с последовательностью MTS (MTS-COX8-OPT, MTS-COX-8-OPT N, MTS-COX-4-OPT, MTS-COX-8-OPT K, MTS-COX-10-OPT, Таблица 2) объединяли с ДНК с последовательностью ND4opt, полученной, как описано в Примере 1.1., при помощи ПЦР с перекрыванием последовательностей (SOE PCR) с использованием праймеров, приведенных в Таблице 2. Праймеры содержали от 5' к 3'-концу короткую последовательность MTS и последовательность прямого праймера.

Прямые праймеры содержали от 5' к 3'-концу короткую последовательность MTS и последовательность прямого праймера для ND4opt. На первом этапе использовали прямой праймер MTS-COX-8-OPT_N, MTS-COX-8-OPT_K, MTS-COX-10-OPT, MTS-СОХ-4-ОРТ для соответствующего фрагмента MTS и обратный праймер ND4_opt_rev, в качестве матрицы использовали полученную ранее плазмиду pAAV-ND4opt. Программа амплификации: предварительное плавление ДНК при 98°С в течение 30 секунд, 15 циклов амплификации, включающих плавление в течение 10 секунд при 98°С, отжиг в течение 30 секунд при 55°С и элонгацию при 72°С в течение 30 секунд, конечную элонгацию при 72°С в течение 3 минут.
На втором этапе использовали прямой праймер MTS-COX8-BamHI_Fw, MTS-COX-4-OPT-Fw, MTS-COX-10-OPT-Fw для соответствующего фрагмента MTS и обратный праймер ND4_opt_rev, в качестве матрицы использовали очищенный продукт ПЦР 1 этапа. Программа амплификации: предварительное плавление ДНК при 98°С в течение 30 секунд, 30 циклов амплификации, включающих плавление в течение 10 секунд при 98°С, отжиг в течение 30 секунд при 55°С и элонгацию при 72°С в течение 30 секунд, конечную элонгацию при 72°С в течение 3 минут.
Полученные в результате амплификации фрагменты MTS-COX-4-ND4, MTS-COX-10-ND4, MTS-COX-8K-ND4 и MTS-COX-8N-ND4 встраивали в вектор pAAV-CMV-MCS. Реакцию рестрикции проводили по методике, рекомендуемой производителем pAAV-CMV-MCS с использованием эндонуклеаз рестрикции BamHI и HindIII (New England Biolabs Inc., США). Реакцию лигирования наработанных вставок и вектора проводили согласно рекомендованному протоколу производителя в объеме 20 мкл. Для лигирования использовали 10Х Т4 DNA Ligation Buffer и Т4 DNA Ligase (New England Biolabs Inc., США), реакцию проводили при 16°С в течение 16 часов (ночи). Инактивирование фермента проводили при 65°С в течение 10 минут.
Трансформацию клеток проводили по стандартному протоколу [Маниатис Т., Фрич Э., Сэмбрук Дж. Методы генетической инженерии. Молекулярное клонирование. - М.: Мир, 1984. - 239-241 с.]. Для проведения трансформации аликвоту химически компетентных клеток Escherichia coli DH5a извлекали из морозильной камеры (-80°С) и помещали в лед для медленного оттаивания. К оттаявшим клеткам добавляли по 5 мкл охлажденной лигазной смеси и инкубировали во льду 30 мин. Проводили тепловой шок в течение 30 сек при 42°С на водяной бане, затем перемещали пробирку с клетками в лед и инкубировали 2 мин. Затем добавляли к клеткам по 1 мл предварительно нагретой до 37°С питательной среды SOC и инкубировали в термостате в течение 1 часа при 37°С и перемешивании со скоростью 180-200 об/мин. Высевали суспензию трансформированных клеток на предварительно подсушенные в термостате при 37°С чашки Петри с LB-агаром и ампициллином по 100 мкл на чашку. Культивировали клетки в термостате при 37°С в течение 16-18 часов.
Для анализа колоний трансформированных клеток проводили ПЦР-скрининг с использованием вектор-специфических праймеров pAAV_For_seq2 и pAAV_Rev_seq2 (Таблица 2) и готовой смеси для ПЦР ScreenMix (Евроген, Россия). Реакционную смесь готовили согласно рекомендациям производителя. В качестве матрицы использовали термолизаты отдельных колоний бактерий. Для этого клетки каждой из 8 колоний отбирали с поверхности питательной среды уколом наконечника микропипетки и переносили в пробирку с 20 мкл воды. Пробирки с суспензией бактерий нагревали при 95°С в течение 5 минут. В ПЦР брали по 2 мкл суспензии. Проводили ПЦР со следующими параметрами: предварительное плавление ДНК при 95°С в течение 3 минут, 25 циклов амплификации, включающих плавление в течение 20 секунд при 95°С, отжиг в течение 20 секунд при 55°С и элонгацию при 72°С в течение 2 минут, конечную элонгацию при 72°С в течение 5 минут. Клоны, для которых методом ПЦР было подтверждено наличие вставки корректного размера, передавали на секвенирование. Отбирали клоны с корректной последовательностью по результатам секвенирования.
В результате культивирования отобранных клонов получили рекомбинантные плазмидные экспрессионные векторы, каждый из которых содержал последовательность ND4opt и последовательность нуклеотидов, кодирующую один из четырех различных вариантов MTS (Фиг. 3-6). Характеристики полученных векторов представлены ниже.
Рекомбинантный плазмидный экспрессионный вектор pAAV-MTS4-ND4opt, экспрессирующий кодонно оптимизированную последовательность гена MT-ND4 (ND4opt, Seq Id No.:1) с последовательностью нуклеотидов, кодирующей MTS СОХ4, содержит в соответствии с физической и генетической картой, представленной на Фиг. 3, следующие элементы:
- участок начала репликации ori;
- левый инвертированный концевой повтор ITR;
- цитомегаловирусный промотор CMV promoter;
- последовательность интрона гена b-глобина человека;
- последовательность SEQ Id No.:3, кодирующую MTS СОХ4 (SEQ Id No.:4)
- последовательность ND4opt Seq Id No.: 1;
- последовательность сигнала полиаденилирования poly(A);
- правый инвертированный концевой повтор ITR;
- промотор гена устойчивости к ампициллину AmpR promoter;
- ген устойчивости к антибиотику ампициллину AmpR.
Рекомбинантный плазмидный экспрессионный вектор pAAV-MTS8-ND4opt, экспрессирующий кодонно оптимизированную последовательность гена MT-ND4 (ND4opt, Seq Id No.:1) с последовательностью нуклеотидов, кодирующей MTS СОХ8, содержит в соответствии с физической и генетической картой, представленной на Фиг. 4, следующие элементы:
- участок начала репликации ori;
- левый инвертированный концевой повтор ITR;
- цитомегаловирусный промотор CMV promoter;
- последовательность интрона гена b-глобина человека;
- последовательность Seq Id No.:5, кодирующую MTS СОХ8 (Seq Id No.:6);
- последовательность ND4opt (Seq Id No.: 1);
- последовательность сигнала полиаденилирования poly(A);
- правый инвертированный концевой повтор ITR;
- промотор гена устойчивости к ампициллину AmpR promoter;
- ген устойчивости к антибиотику ампициллину AmpR.
Рекомбинантный плазмидный экспрессионный вектор pAAV-MTS8(N)-ND4opt, экспрессирующий кодонно оптимизированную последовательность гена MT-ND4 (ND4opt, Seq Id No.:1) с последовательностью нуклеотидов, кодирующей MTS COX8(N), содержит в соответствии с физической и генетической картой, представленной на Фиг. 5, следующие элементы:
- участок начала репликации ori;
- левый инвертированный концевой повтор ITR;
- цитомегаловирусный промотор CMV promoter;
- последовательность интрона гена b-глобина человека;
- последовательность SEQ Id No.:7, кодирующую MTS COX8(N) (SEQ Id No.: 8);
- последовательность ND4opt Seq Id No.: 1;
- последовательность сигнала полиаденилирования poly(A);
- правый инвертированный концевой повтор ITR;
- промотор гена устойчивости к ампициллину AmpR promoter;
- ген устойчивости к антибиотику ампициллину AmpR.
Рекомбинантный плазмидный экспрессионный вектор pAAV-MTS10-ND4opt, экспрессирующий кодонно оптимизированную последовательность гена MT-ND4 (ND4opt, Seq Id No.:1) с последовательностью нуклеотидов, кодирующей MTS СОХ10, содержит в соответствии с физической и генетической картой, представленной на Фиг. 6, следующие элементы:
- участок начала репликации ori;
- левый инвертированный концевой повтор ITR;
- цитомегаловирусный промотор CMV promoter;
- последовательность интрона гена b-глобина человека;
- последовательность SEQ Id No.:9, кодирующую MTS СОХ10 (SEQ Id No.: 10);
- последовательность ND4opt Seq Id No.: 1;
- последовательность сигнала полиаденилирования poly(A);
- правый инвертированный концевой повтор ITR;
- промотор гена устойчивости к ампициллину AmpR promoter;
- ген устойчивости к антибиотику ампициллину AmpR.
Пример 1.4. Оценка влияния экспрессии и коэкспрессии генов после трансфекции HEK293
Так как мутации в генах, кодирующих компоненты электрон-транспортной цепи, могут приводить к повышению концентрации ионов кальция и увеличению продукции АФК в митохондриях, для оценки активности полученных в Примере 1.3. экспрессионных векторов использовали 3 сенсора, которые флуоресцируют в зеленой области спектра.
1) pCase12-Mito (cat#FP992, Евроген, Россия) генетически кодируемый сенсор для измерения концентрации кальция в митохондриях;
2) pHyper-dMito - генетически кодируемый сенсор для измерения АФК внутри митохондрий (cat#FP942, Евроген, Россия);
3) краситель H2DCFDA для измерения количества внутриклеточных АФК.
Существует несколько методов детекции АФК, в том числе Н2О2.
К прямым методам детекции относится методика с использованием клеточно-проницаемого флуорогенного зонда H2DFCDA (2',7'-Дихлоро-дигидро-флуоресцеин диацетат). Будучи клеточно-проницаемым нефлуоресцентный H2DFCDA быстро диффундирует в клетки, где позже деацетилируется клеточными эстеразами и окисляется АФК до дихлорфлуоресцеина (DFC). DFC характеризуется яркой флуоресценцией в зеленой области спектраи может выявляться при микроскопии, титровании в микропланшетах и цитометрии (ex 485/em 535). Интенсивность флуоресценции пропорциональна уровню АФК в клетке [Woolley J.F., Stanicka J., Cotter T.G. Recent advances in reactive oxygen species measurement in biological systems //Trends in biochemical sciences. 2013. T. 38. №. 11. C. 556-565].
Другой подход использует генетически кодируемые биосенсоры, позволяющие проводить анализ изменений перекиси водорода - одной из основных АФК, образующихся в клетке. В таких биосенсорах может быть использован, например, чувствительный к восстановлению и окислению зеленый флуоресцентный белок (roGFP), который может существовать в двух различных состояниях окисления: восстановленный дитиол или окисленный дисульфид, каждое из которых обладает различными флуоресцентными свойствами [Hanson G.Т. et al. Investigating mitochondrial redox potential with redox-sensitive green fluorescent protein indicators //Journal of Biological Chemistry. 2004. T. 279. №. 13. C. 13044-13053].
Группой Всеволода Белоусова в Институте Биоорганической Химии им. Шемякина и Овчинникова в 2006 году был разработан генетически кодируемый биосенсор на основе слитого белка - HyPer, - высокоспецифичный флуоресцентный биосенсор для обнаружения Н2О2 внутри живых клеток. HyPer состоит из циклически пермутированного желтого флуоресцентного белка (cpYFP), встроенного в регуляторный домен прокариотического Н2О2-чувствительного белка, OxyR. В отличие от обычно используемых производных дихлорфлуоресцеина (DCF), HyPer обладает высокой специфичностью к Н2О2, не продуцирует АФК под действием света и не чувствителен к супероксиду, окисленному глутатиону, оксиду азота и пероксинитриту [Belousov V.V. et al. Genetically encoded fluorescent indicator for intracellular hydrogen peroxide //Nature methods. - 2006. T. 3. №. 4. - C. 281-286].
Для экспериментальной работы использовали вектор pHyPer-dMito (cat#FP942, Евроген), предназначенный для экспрессии HyPer, слитого с MTS, под контролем конститутивного CMV-промотора в клетках млекопитающих.
Повреждение митохондрий вызывает снижение продукции АТФ, что приводит к открытию митохондриальной Са2+ -зависимой поры (mPTP - Mitochondrial Permeability Transition pore). Митохондриальная пора (mPTP) - неселективный канал, который играет значительную роль в кальциевом обмене между митохондриями и средой. Открытие митохондриальной поры влияет на ионный гомеостаз клетки, скорость образования активных форм кислорода и активацию клеточной гибели.
На данный момент разработаны флуоресцентные кальциевые зонды на основе хелатора ВАРТА, флуоресценция которых зависит от концентрации ионов кальция в среде (Fluo, Oregon Green, Fura Red). Однако для количественного определения могут быть использованы только возбуждаемые ультрафиолетом Fura 2 и Indo.
В 2007 году был разработан генетически кодируемый флуоресцентный сенсор ионов Са2+ Case12, который имеет высокий динамический диапазон для прямого измерения изменений внутриклеточного Са2+ при различных физиологических и патологических состояниях [Souslova Е.A. et al. Single fluorescent protein-based Ca2+ sensors with increased dynamic range //BMC biotechnology. 2007. T. 7. №. 1. C. 1-10]. Данный сенсор чувствителен к изменению концентрации кальция в физиологическом диапазоне от сотен наномолей до микромолярных концентраций. Связывание Са2+ быстро и обратимо, что позволяет отслеживать высокочастотные колебания данных ионов. В ответ на повышение концентрации Са2+ наблюдается увеличение интенсивности флуоресценции до 12 раз. Флуоресценция Case12 характеризуется одиночными максимумами возбуждения/испускания с максимумом при 491/516 нм.
Для экспериментальной работы нами был использован вектор pCase12-dMito, предназначенный для экспрессии под контролем конститутивного CMV-промотора в клетках млекопитающих Case12, слитого с последовательностью, кодирующей пептид митохондриальной локализации (cat#FP992, Евроген, Россия).
Для того чтобы оценить влияние плазмид, экспрессирующих ND4opt с последовательностью, кодирующей один из вариантов MTS (Фиг. 3-6) на количество АФК, клетки линии HEK293TN (Bio-Cat, Германия) одновременно трансфицировали 2 плазмидами:
1) сенсором pHyper-dMito
2) одним из вариантов плазмид, экспрессирующих ND4opt с последовательностью, кодирующей один из вариантов MTS, или плазмидой pAAV-MCS, не содержащей трансген.
Для приготовления трансфекционной смеси в пробирку добавляли ДНК в расчете 250 нг ДНК на лунку 48-луночного планшета для 80 тысяч клеток. В другую пробирку добавляли реагент для трансфекции полиэтиленимин (PEI-MAX, MW 40000, Polysciences, США) из расчета массового соотношения ДНК:PEI 1:5. К ДНК и PEI добавляли среду OptiMEM до объема из расчета 15 мкл каждого реагента для каждой лунки. Содержимое двух пробирок объединяли и инкубировали 10-15 минут. В каждую лунку 48-луночного планшета вносили по 30 мкл трансфекционной смеси и сразу добавляли по 300 мкл клеточной суспензии с 80 тысячами клеток в среде ДМЕМ с добавлением 4,5 г/л глюкозы и 2% об. эмбриональной бычьей сыворотки (FBS). Инкубировали при 37°С, 5% СО2. Питательную среду заменяли через 24 часа на среду ДМЕМ, содержащую 10% об. FBS в объеме 300 мкл на лунку. После завершения этапа трансфекции культуральный планшет переносили в систему для прижизненного анализа клеток IncuCyte S3 со следующими настройками:
- В системе был выбран тип сканирования по расписанию (Scan on schedule).
- В поле Create or Restore Vessel соответствующий тип используемого сосуда и каналы для съемки (Brightfield и Green).
- В поле Vessel Location было выбрано положение в системе, соответствующее положению сканируемого планшета.
- В поле Scan Pattern были выбраны анализируемые лунки планшета и количество микрофотографий на каждую лунку планшета (9).
- В поле Vessel Notebook было введено название эксперимента в системе, а также клеточный тип.
- В поле Analysis Setup выбраны следующие настройки: Basic Analyzer, GFP per cell.
- В поле Scan Schedule было сформировано расписание сканирований для анализируемых лунок планшета.
Полученные результаты продемонстрировали негативное влияние процесса трансфекции двумя плазмидами на состояние клеток и количество АФК, в то время как снижение генетической нагрузки (трансфекция только одной плазмидой pHyper-Mito) не приводило к высоким значениям сигнала сенсора.
При этом экспрессия ND4opt с последовательностью, кодирующей любой из различных вариантов MTS, компенсировала негативный эффект трансфекции двумя плазмидами, снижая уровень митохондриальных АФК в клетках HEK293 по абсолютному значению сигнала сенсора в 2 и более раз по сравнению с клетками, трансфицированными плазмидой pHyper-Mito и плазмидой pAAV-MCS, не экспрессирующей трансген. Наибольший уровень снижения количества АФК наблюдали в случае использования плазмиды pAAV-MTS8-ND4opt - 57,85% (Фиг. 7).
Пример 1.5. Оценка влияния экспрессии ND4opt на количество активных форм кислорода с помощью биосенсора pHyper-dMito на модели первичных фибробластов клеточной модели LHON.
В качестве релевантной клеточной модели LHON использовали первичные фибробласты из биобанка ФГБНУ «Медико-генетический научный центр имени академика Н.П. Бочкова», несущие наиболее часто встречающуюся мутацию m. 11778G>A в гене MT-ND4. Наличие мутации подтвердили секвенированием по Сенгеру. Клетки первичных дермальных фибробластов культивировали в среде ДМЕМ с высоким содержанием глюкозы (4,5 г/л) и 10% эмбриональной бычьей сыворотки, содержащей L-Gln и заменимые аминокислоты. Среду культивирования меняли каждые 2-3 дня. После достижения клетками 80-90% конфлюентности проводили замораживание клеток с целью формирования рабочего банка клеточных линий. Для этого трипсинизировали клетки 0,25% раствором трипсина-ЭДТА, отмывали от излишков трипсина в фосфатно-солевом буфере, центрифугировали при 200 g, разводили промытые клетки в эмбриональной бычьей сыворотке с 10% ДМСО, переносили аликвоты клеточных суспензий в криоампулы в количестве 1 млн клеток на ампулу, помещали ампулы в морозильную камеру с температурой -80°С. После замораживания криоампулы с клетками переносили в криохранилище с жидким азотом для хранения. В дальнейшем клетки размораживали по мере необходимости по стандартным протоколам.
Клетки трансфицировали одновременно двумя плазмидами: pHyper-dMito и одной из плазмид, экспрессирующих ND4opt с последовательностью, кодирующей один из вариантов MTS (Фиг. 3-6).
Для трансфекции фибробластов готовили трансфекционную смесь. Для этого в центрифужную пробирку добавляли плазмидную ДНК из расчета 250 нг ДНК на лунку 48-луночного планшета. В другую пробирку добавляли реагент для трансфекции GenJect 39 (GenJect 39, Molecta, Россия) из расчета массового соотношения ДНК: GenJect 39 1:2. К ДНК и GenJect 39 добавляли среду ДМЕМ до общего объема из расчета 15 мкл для каждой лунки 48-луночного планшета. Для получения трансфекционной смеси содержимое двух пробирок объединяли и инкубировали 10-15 минут. К осадку клеток, полученному после центрифугирования при 200g, из расчета 80 тысяч клеток на лунку, добавляли трансфекционную смесь объемом из расчета 30 мкл на лунку и инкубировали 20 минут при 37°С. К клеточной суспензии добавляли среду ДМЕМ с 10% об. FBS из расчета 300 мкл на лунку. В лунки 48-луночного планшета переносили по 350 мкл полученной клеточной суспензии и инкубировали при 37°С, 5% СО2. Питательную среду заменяли через 24 часа на среду ДМЕМ объемом 300 мкл, содержащую 10% об. FBS. После завершения этапа трансфекции переносили культуральный планшет в систему для прижизненного анализа клеток IncuCyte S3 с настройками, описанными ранее в Примере 1.4.
В результате трансфекции клеток плазмидным экспрессионным вектором pAAV-MTS8-ND4opt, экспрессирующим ND4opt с последовательностью, кодирующей MTS СОХ8, наблюдается значительное снижение уровня АФК: на 50% по сравнению с контролем, подвергшимся трансфекции плазмидным экспрессионным вектором pAAV-MCS (Фиг. 8).
Пример 1.6. Оценка влияния экспрессии ND4opt на митохондриальный уровень кальция с помощью биосенсора pCase12-Mito на модели первичных фибробластов клеточной модели LHON
С использованием генетически кодируемого сенсора pCase12-Mito возможно определение митохондриальной концентрации ионов кальция: жизненно важного медиатора энергетического метаболизма митохондрий и запуска процессов клеточной гибели.
Клетки фибробластов, несущих мутацию m.11778G>A в гене MT-ND4, получали, как описано выше в Примере 1.5. Клетки трансфицировали одновременно двумя плазмидами: pCase12-Mito и одной из плазмид, экспрессирующих ND4opt с последовательностью, кодирующей один из вариантов MTS (Фиг. 3-6), как описано выше.
Детекцию флуоресценции сенсора кальция pCase12-Mito проводили с использованием системы для прижизненного анализа клеток IncuCyte S3 (условия эксперимента аналогичны описанным ранее в Примере 1.4.).
В результате трансфекции плазмидным экспрессионным вектором pAAV-MTS8-ND4opt, экспрессирующим ND4opt с последовательностью, кодирующей MTS СОХ8, наблюдается снижение уровня катионов Са2+ в митохондриях на ~50% (Фиг. 9).
Пример 1.7. Оценка влияния экспрессии ND4opt на количество активных форм кислорода с помощью красителя на модели первичных фибробластов клеточной модели LHON
Как было описано выше, мутации в гене MT-ND4 способствуют повышению количества АФК в клетках и при восстановлении функции этого гена наблюдается понижение количества АФК. В связи с этим мы использовали метод оценки уровня АФК для проверки функциональной активности последовательности ND4opt в сочетании с последовательностью, кодирующей один из вариантов MTS, при введении в клетки в составе плазмидного экспрессионного вектора pAAV-MTS-ND4opt. Для оценки количества АФК использовали 2',7'-дихлородигидрофлуоресцеин диацетат (H2DCFDA) как один из широко используемых реагентов для измерения количества АФК в живых клетках. H2DCFDA представляет собой нефлуоресцирующее производное флуоресцеина - его восстановленную ацетилированную форму. Реагент начинает флуоресцировать только после отщепления ацетильных групп и окисления внутри клетки под воздействием АФК, превращаясь в 2',7'-дихлорофлуоресцеин (DCF). Чем выше уровень АФК в клетках, тем больше накапливается флуоресцирующий продукт реакции, который можно оценивать с помощью планшетных анализаторов в определенной области спектра.
Таким образом, для определения функциональной активности ND4opt с одной из последовательностей, кодирующих MTS, мы проводили трансфекцию полученными экспрессионными векторами первичных фибробластов с дефектным геном MT-ND4, после чего измеряли уровень АФК в клетках.
Клеточные культуры фибробластов трансфицировали плазмидными экспрессионными векторами, описанными в Примере 1.2., способом, описанным ранее в Примере 1.5. Далее культуральный планшет культивировали при 37°С и 5% СО2 в течение 7 суток. Действия с красителем проводили в темноте. Клеточный монослой в лунках 48-луночного планшета дважды промывали 250 мкл фосфатно-солевого буфера (PBS). Удаляли остаток жидкости и вносили 237,5 мкл PBS.
В каждую лунку вносили по 12,5 мкл раствора красителя H2DCFDA до конечной концентрации 5 мкМ. Инкубировали планшет при 37°С в течение 2 часов.
После завершения инкубации с красителем H2DCFDA культуральный планшет переносили в систему ClarioStar-Plus. В системе выбирали следующий тип сканирования: измерение при максимуме поглощения при λ 483 нм, максимум флуоресценции при λ 530 нм. После измерения клеточный монослой трипсинизировали и производили подсчет клеток на счетчике Countess II по протоколу производителя.
Результаты эксперимента (Фиг. 10) показали, что экспрессия последовательности по настоящему изобретению ND4opt способна компенсировать вклад трансфекции в избыточное накопление АФК, а также снижать уровень АФК в 2 и более раз при длительной инкубации (7 суток) по сравнению с фибробластами, трансфицированными плазмидой pAAV-MCS без ND4opt (эквивалентная генетическая нагрузка для моделирования условий стресса).
Было показано, что трансфекция первичных фибробластов клеточной модели LHON плазмидными экспрессионными векторами, экспрессирующими ND4opt с одной из последовательностей, кодирующих MTS, приводит к снижению уровня флуоресценции в клетках, что свидетельствует о снижении АФК в клетках, по сравнению с нетрансфицированными клетками и клетками, трансфицированными исходным плазмидным экспрессионным вектором pAAV-MCS, не содержащим последовательность ND4opt и последовательность нуклеотидов, кодирующую MTS. Из исследованных вариантов плазмидных экспрессионных векторов, экспрессирующих ND4opt с последовательностью нуклеотидов, кодирующей MTS, больше всего способствовал снижению уровня АФК вариант pAAV-MTS8-ND4opt (Фиг. 10). Использование вектора pAAV-MTS8-ND4opt привело к снижению уровня флуоресценции, обусловленной АФК, не менее чем на 55% и 40,2%, вектора pAAV-MTS8(N)-ND4opt - на 35% и 13,2%, вектора pAAV-MTS10-ND4opt - к снижению на 21% и увеличению на 5,1% и вектора pAAV-MTS4-ND4opt - к снижению на 23% и увеличению на 3,2% относительно клеток, трансфицированных исходным вектором pAAV-MCS и нетрансфицированных клеток соответственно.
Пример 1.8. Оценка влияния экспрессии последовательности ND4opt на маркеры окислительного стресса после трансдукции в составе вирусных векторов
Для получения вирусных AAV векторов использовали плазмидные экспрессионные векторы pAAV-MTS8-ND4opt, pAAV-MTS10-ND4opt, а также коммерчески доступные хелперную плазмиду (pHelper) и упаковочную плазмиду для AAV6 pAAV-RC6 (cat#VPK-426, Cell Biolabs Inc, США).
Клетки суспензионной клеточной линии HEK293 размораживали и культивировали согласно стандартным операционным процедурам и методикам. На момент получения расчетная плотность клеточной культуры составила 106 кл/мл, объем клеточной культуры 30 мл. Трансфекцию проводили в колбах Эрленмейера объемом 500 мл (рабочий объем 175 мл). Посевная доза 87,5⋅106 клеток. Трансфекцию плазмидами проводили через 12 часов после засева. Условия трансфекции: концентрация клеток при трансфекции 106 кл/мл, жизнеспособность 85-90%, масса ДНК 1,5 мкг/1 млн клеточной биомассы. Соотношение ДНК:PEI 1:5, объем трансфицирующей смеси 5% от объема клеточной культуры. После трансфекции клетки инкубировали при 37°С, 5%СО2, 75% влажности, 100 об/мин в течение 120 ч.
Для лизиса клеток в колбу добавляли Твин-20 до концентрации 0,05% и инкубировали в течение 1 часа). Далее добавляли эндонуклеазу (Диа-М, Россия) до 20 МЕ/мл и MgCl2 до 1-2 мМ и инкубировали в течение 1 часа. Центрифугировали лизаты в течение 10 мин при 3000 g и фильтровали через фильтры с размером пор 0,22 мкм. Проводили концентрирование фильтратов методом тангенциальной фильтрации с отсечением молекул размером больше 100 кДа, из 525 мл в 50 мл, при трансмембранном давлении 1,5-2 бар. Аффинную хроматографию проводили на сорбенте AAVX (Thermo Fisher Scientific, США) в соответствии с рекомендациями производителя.
Оценивали вирусный титр полученных образцов AAV с помощью ПЦР в режиме реального времени (qPCR), а также физический титр и наличие примесей (низкомолекулярных или высокомолекулярных-агрегатов) с помощью метода динамического рассеяния света (DLS). Во всех образцах обнаруживали частицы размером преимущественно 20-25 нм и минорное количество высокомолекулярных примесей (агрегатов).
Фибробласты, полученные как описано в Примере 1.5., трансдуцировали вирусными частицами в дозе 10000 MOI (отношение количества вирусных частиц к количеству клеток-мишеней, или множественность инфекции, от англ. multiplicity of infection). В качестве отрицательного контроля флуоресценции использовали нетрансдуцированные клетки. Анализ флуоресценции проводили в условиях, описанных выше в Примере 1.2.
Было показано, что трансдукция первичных фибробластов клеточной модели LHON вирусными векторами AAV6, экспрессирующими ND4opt с последовательностью, кодирующей MTS СОХ8, приводит к снижению уровня флуоресценции на ~25%, что свидетельствует о снижении АФК в этих клетках (Фиг. 11).
Таким образом, изобретение позволяет восстановить функцию НАДН-дегидрогеназы в клетках, что выражается в снижении активных форм кислорода и концентрации ионов кальция в митохондриях, иными словами, наблюдается снижение последствий нарушения функции митохондрий в клетках с мутацией в гене MT-ND4.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="ND4_listing.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2023-10-17">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023126578</ApplicationNumberText>
<FilingDate>2023-10-17</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Автономная некоммерческая
образовательная организация высшего образования
"Научно-Технологический Университет
"Сириус"</ApplicantName>
<ApplicantNameLatin>Sirius University of Science and
Technology</ApplicantNameLatin>
<InventionTitle languageCode="ru">Последовательность нуклеотидов для
аллотопической экспрессии белка MT-ND4</InventionTitle>
<SequenceTotalQuantity>25</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>1383</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1383</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q55">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgctgaaactgatcgtgccaacaattatgctgctgccactgacctggc
tgagcaagaagcacatgatctggatcaacaccaccacccacagcctgatcatcagcatcatccccctgct
ctttttcaaccagatcaacaacaacctgttcagctgcagccctaccttctccagcgaccccctgaccaca
cctctgctgatgctgacaacatggctgctgcctctgaccatcatggccagccaaagacacctgtcaagcg
agcctctgagcagaaagaagctgtatctgagcatgctgatctccctgcaaatcagcctcatcatgacctt
taccgccaccgagctgatcatgttctacatcttcttcgagacaaccctcattcctaccctggccatcatc
accagatggggcaaccagcctgagaggctgaacgccggtacatacttcctgttttacacacttgtgggca
gcctgcctctgctaatcgccctgatctacacccataataccctgggatctcttaacatcctgctgctgac
gcttacagcccaggagctgagcaacagctgggccaacaacctgatgtggctggcctacaccatggctttc
atggtcaagatgcctctgtacggcctgcacctgtggctgcccaaggcccacgtggaagcccctatcgccg
gcagcatggtgctggccgccgtgctgctcaagctgggcggctacggcatgatgcggctgaccctgatcct
gaatcctctgactaagcacatggcctaccccttcctggtgctgagcctgtggggaatgatcatgacatct
agcatctgtctgagacagactgatctgaagagcctgatcgcctattcttctatcagccacatggctctgg
tggtgaccgctatcctgattcagaccccttggtcctttaccggcgctgtgatcctgatgatcgcacacgg
cctgaccagcagcctgctgttctgcctggctaattctaattacgagagaacacatagccggatcatgatc
ctgtcccagggcctgcagacccttctgcctctgatggccttctggtggctgctggcctctctcgccaacc
tggctctgccacctaccattaacctgctgggcgaactgtccgtgttagttaccacattcagctggagcaa
catcaccctgttactgaccggcctgaatatgctggtgaccgccctctacagcctgtacatgttcaccacc
acccagtggggctctctgacgcaccacatcaacaacatgaaacccagcttcacacgggaaaacacactga
tgttcatgcacctgtctcccatcctgctgctgtccctgaaccccgacatcatcacaggatttagctccta
atga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>459</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..459</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q56">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLKLIVPTIMLLPLTWLSKKHMIWINTTTHSLIISIIPLLFFNQINNNL
FSCSPTFSSDPLTTPLLMLTTWLLPLTIMASQRHLSSEPLSRKKLYLSMLISLQISLIMTFTATELIMFY
IFFETTLIPTLAIITRWGNQPERLNAGTYFLFYTLVGSLPLLIALIYTHNTLGSLNILLLTLTAQELSNS
WANNLMWLAYTMAFMVKMPLYGLHLWLPKAHVEAPIAGSMVLAAVLLKLGGYGMMRLTLILNPLTKHMAY
PFLVLSLWGMIMTSSICLRQTDLKSLIAYSSISHMALVVTAILIQTPWSFTGAVILMIAHGLTSSLLFCL
ANSNYERTHSRIMILSQGLQTLLPLMAFWWLLASLANLALPPTINLLGELSVLVTTFSWSNITLLLTGLN
MLVTALYSLYMFTTTQWGSLTHHINNMKPSFTRENTLMFMHLSPILLLSLNPDIITGFSS</INSDSeq_
sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>66</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..66</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q59">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgttggctaccagggtatttagcctagttggcaagcgagcaatttcca
cctctgtgtgtgtacga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q60">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLATRVFSLVGKRAISTSVCVR</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>75</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..75</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q61">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtccgtcctgacgccgctgctgctgcggggcttgacaggctcggccc
ggcggctcccagtgccgcgcgccaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q62">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSVLTPLLLRGLTGSARRLPVPRAK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>75</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..75</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q63">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtccgtcctgacgccgctgctgctgcggggcttgacaggctcggccc
ggcggctcccagtgccgcgcgccaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q64">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSVLTPLLLRGLTGSARRLPVPRAN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>84</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..84</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q65">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggccgcatctccgcacactctctcctcacgcctcctgacaggttgcg
taggaggctctgtctggtatcttgaaagaagaact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q66">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MAASPHTLSSRLLTGCVGGSVWYLERRT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>108</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q70">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggccgccatgcgctggcgatggtggcagcggctgttaccttggaggt
tgctgcaggcccgtggctttccacaaaattctgcacccagcctgggcgcccgcacctac</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q69">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MAAMRWRWWQRLLPWRLLQARGFPQNSAPGLGARTY</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatatggatccatgtccgtcctga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>107</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..107</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatatggatccatgtccgtcctgacgccgctgctgctgcggggcttga
caggctcggcccggcggctcccagtgccgcgcgccaacctgaaactgatcgtgccaac</INSDSeq_se
quence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>98</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..98</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatatggatccatgttggctaccagggtatttagcctagttggcaagc
gagcaatttccacctctgtgtgtgtacgactgaaactgatcgtgccaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>107</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..107</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatatggatccatgtccgtcctgacgccgctgctgctgcggggcttga
caggctcggcccggcggctcccagtgccgcgcgccaagctgaaactgatcgtgccaac</INSDSeq_se
quence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>116</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..116</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatatggatccatggccgcatctccgcacactctctcctcacgcctcc
tgacaggttgcgtaggaggctctgtctggtatcttgaaagaagaactctgaaactgatcgtgccaac</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatatggatccatggccgccatgcgctggcg</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttggcacgatcagtttcaggtaggtgcgggcgcccaggccc</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatatggatccatgttggctaccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatatggatccatggccgcatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>37</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atatataagctttcattaggagctaaatcctgtgatg</INSDSeq_se
quence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttggcacgatcagtttcaggtaggtgcgggcgcccaggccc</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q52">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcttcctcccacagctcctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttccagggccaggagaggca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Пептид митохондриальной локализации, нуклеиновая кислота для аллотопической экспрессии гена MT-ND4, содержащий ее экспрессионный вектор и его применение | 2023 |
|
RU2817420C1 |
| МИНИ-БЕЛОК USH2A, НУКЛЕИНОВАЯ КИСЛОТА, КОДИРУЮЩАЯ МИНИБЕЛОК USH2A, И СОДЕРЖАЩИЙ ЕЕ ЭКСПРЕССИОННЫЙ ВЕКТОР ДЛЯ ГЕННОЙ ТЕРАПИИ | 2023 |
|
RU2822884C1 |
| Нуклеиновая кислота, предназначенная для снижения массы тела млекопитающего, экспрессионный вектор для экспрессии в клетках млекопитающего, способ его доставки и способ снижения массы тела млекопитающего | 2023 |
|
RU2810191C1 |
| Применение вектора для снижения иммунного ответа при вирусной доставке AIPL1 | 2023 |
|
RU2818342C1 |
| Нуклеиновая кислота, содержащая последовательность гена PRDM16, предназначенная для снижения массы тела млекопитающего, экспрессионный вектор для экспрессии в клетках млекопитающего, способ его доставки и способ снижения массы тела млекопитающего | 2024 |
|
RU2834035C1 |
| Кодон-оптимизированная последовательность нуклеотидов, кодирующая hAIPL1, и её содержащий экспрессионный вектор | 2021 |
|
RU2785621C1 |
| АДЕНОАССОЦИИРОВАННЫЙ ВИРУСНЫЙ ВЕКТОР, СОСТОЯЩИЙ ИЗ БЕЛКОВ КАПСИДА РНР.В, НУКЛЕИНОВОЙ КИСЛОТЫ, КОДИРУЮЩЕЙ БЕЛОК SMN, И ЕГО ПРИМЕНЕНИЕ | 2022 |
|
RU2833225C2 |
| Генетическая конструкция и способ экспрессии ERVW-1 и MYMK | 2025 |
|
RU2839754C1 |
| Кодон-оптимизированная нуклеиновая кислота, которая кодирует белок SMN1, и ее применение | 2020 |
|
RU2742837C1 |
| Препарат для стимулирования регенерации и нейропротекции нервной ткани и способ его использования | 2023 |
|
RU2828544C1 |
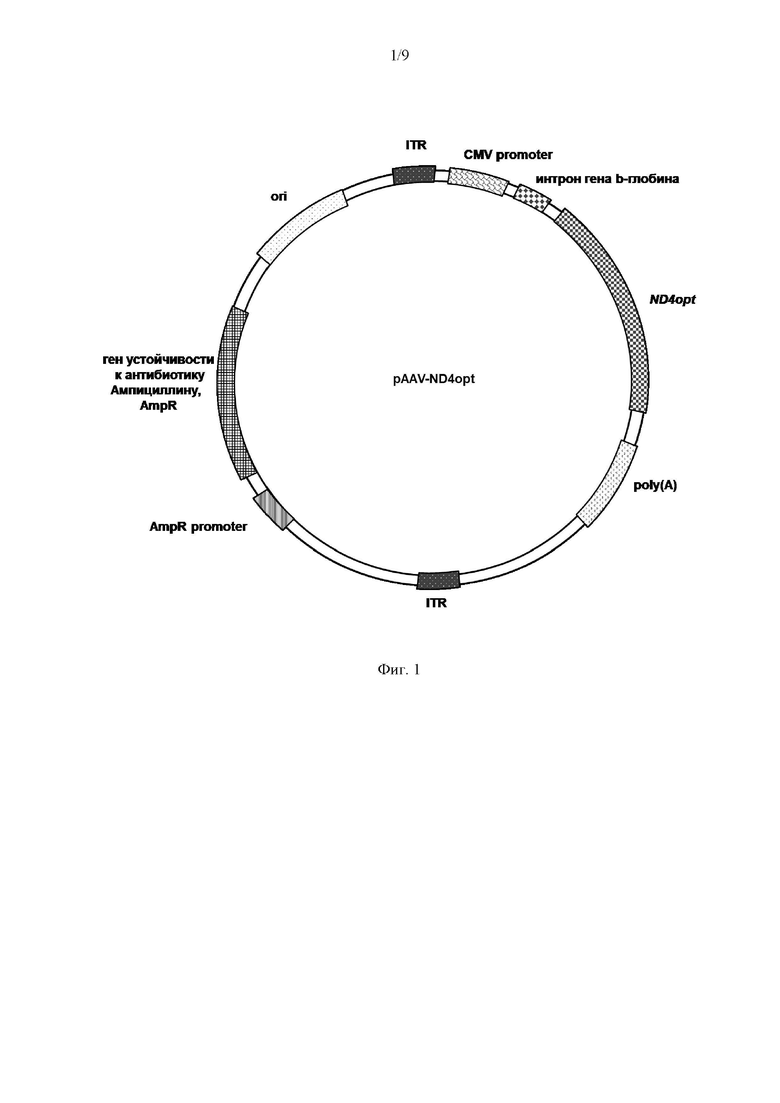

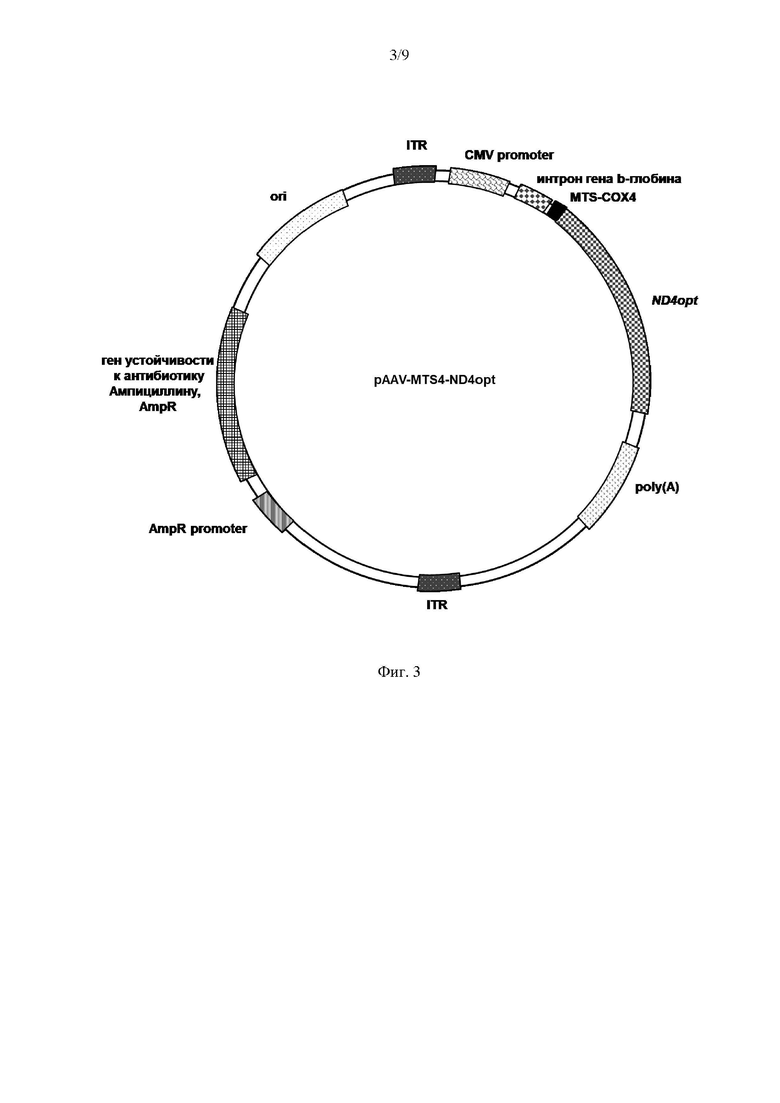
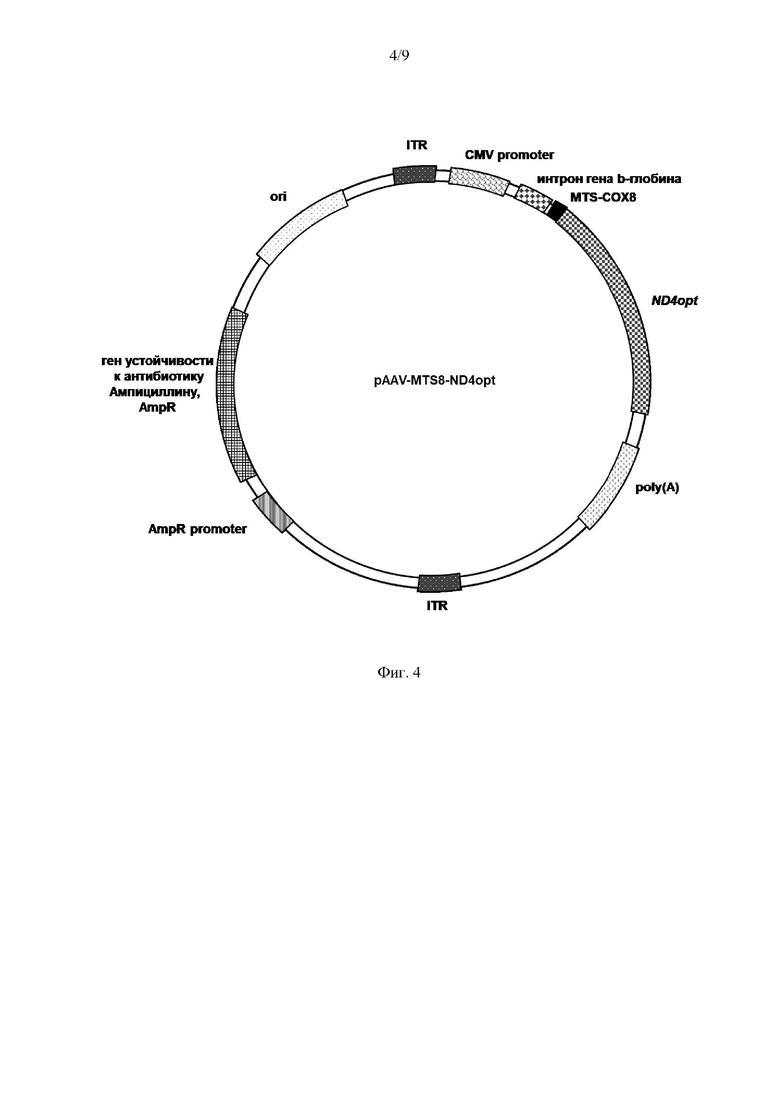
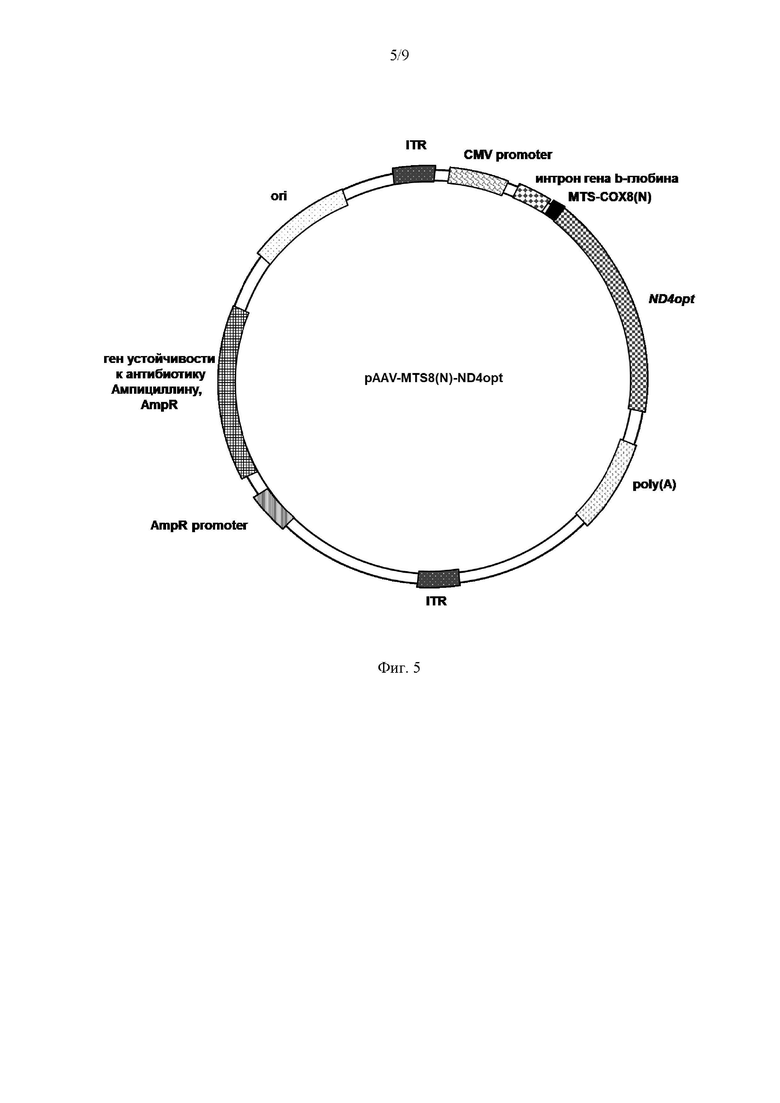
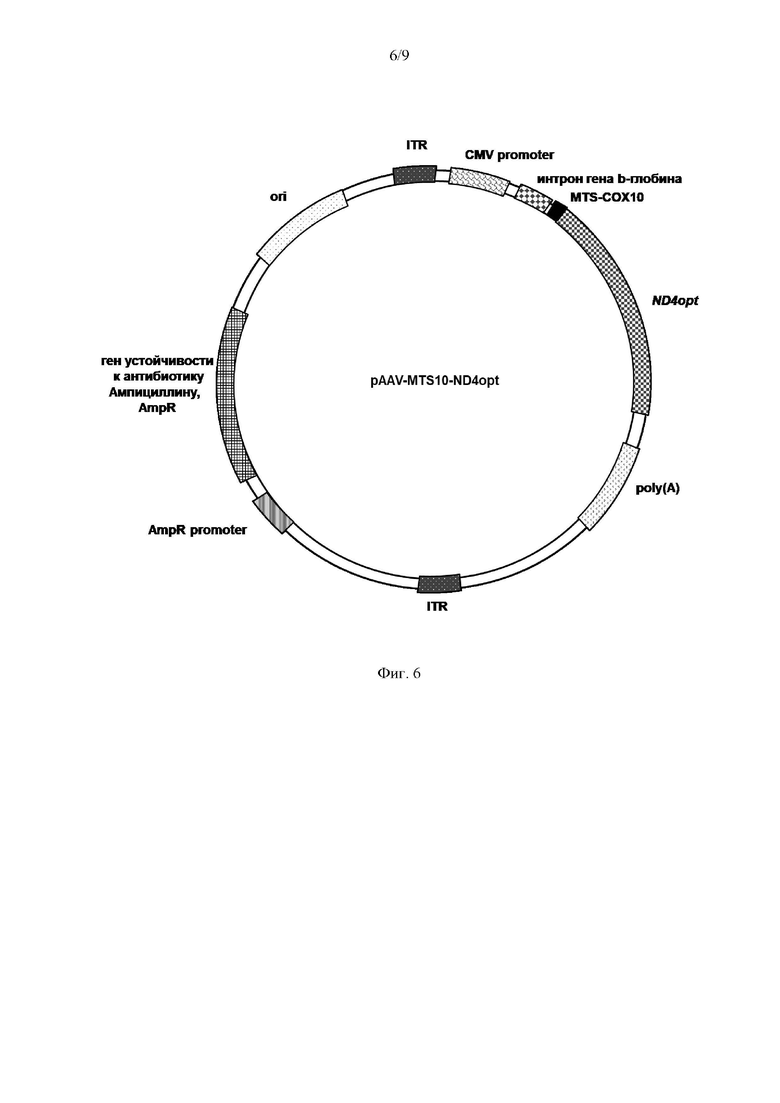
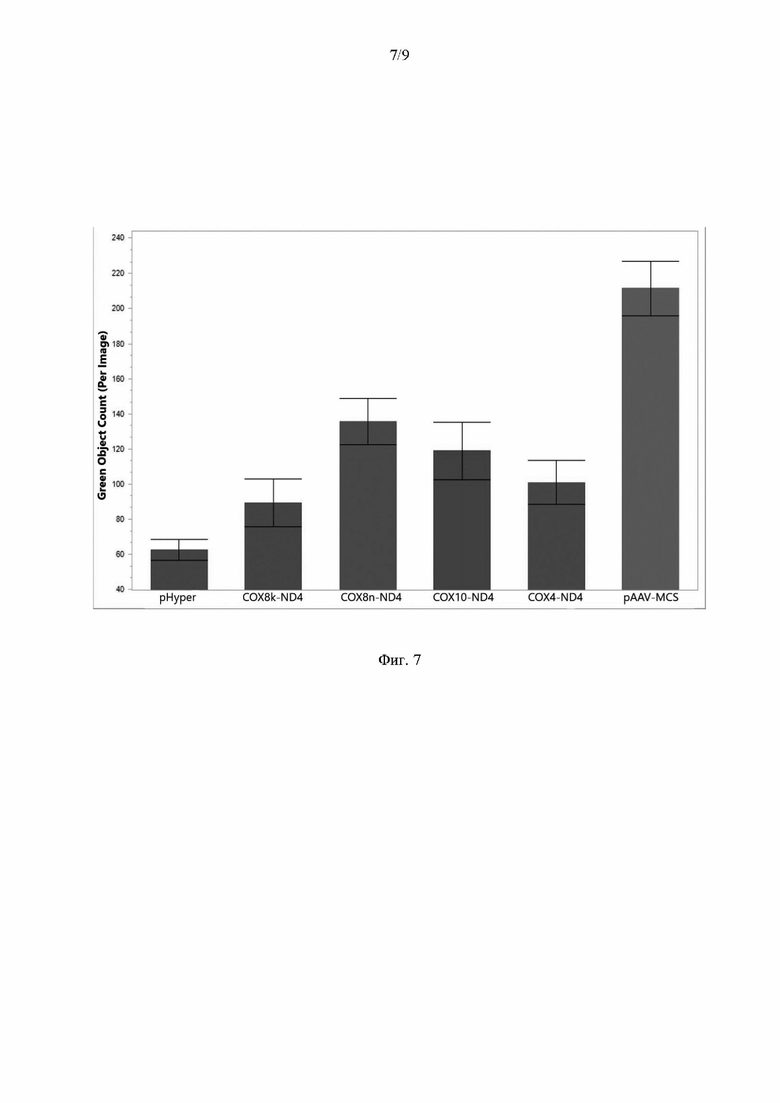
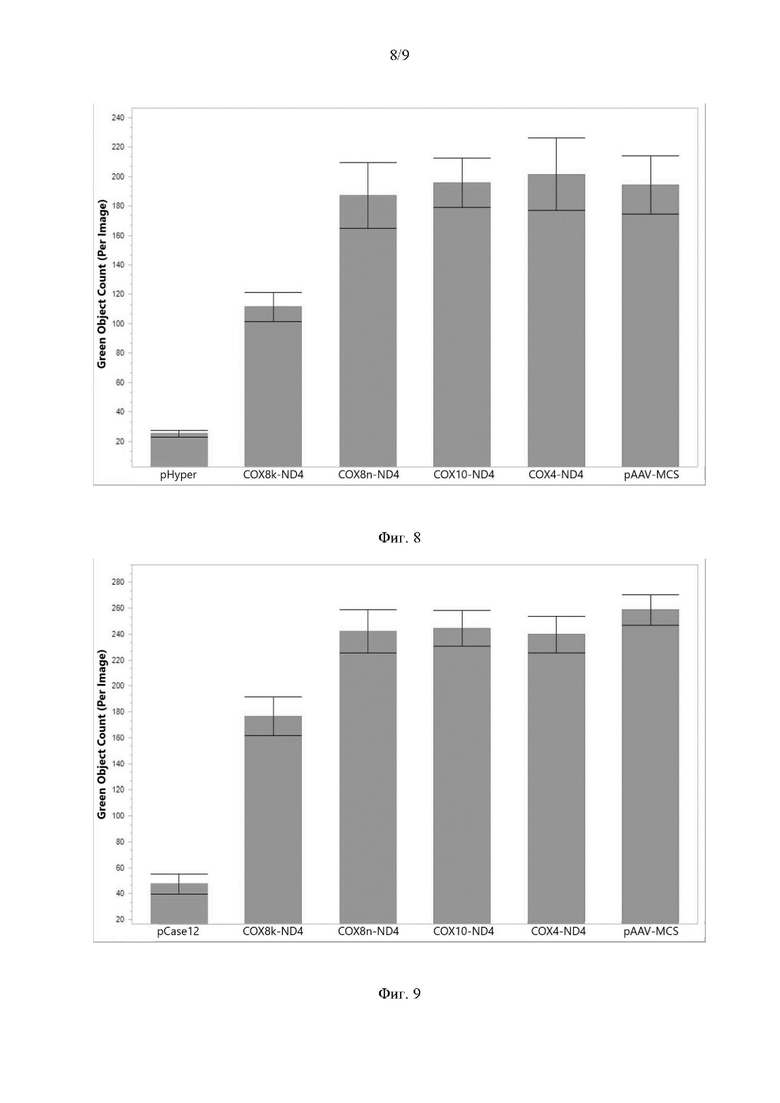
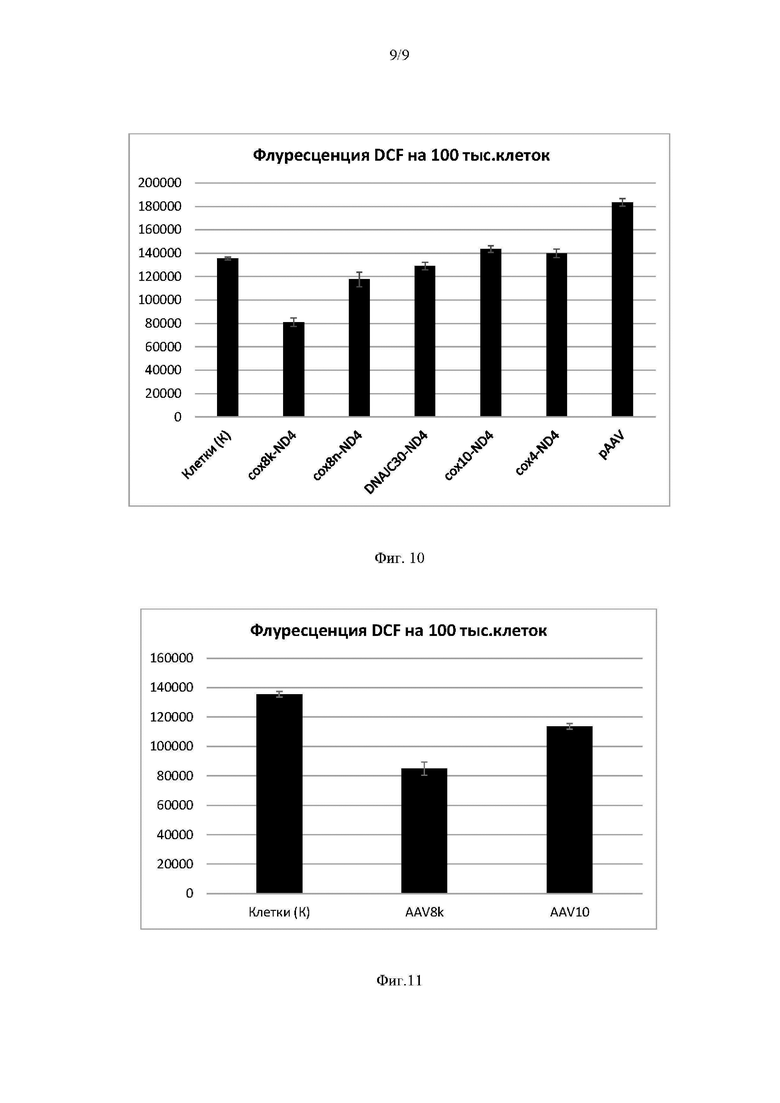
Группа изобретений относится к биотехнологии и молекулярной биологии, в частности генной инженерии. Предложена нуклеиновая кислота для аллотопической экспрессии гена MT-ND4, а также экспрессионный вектор, содержащий упомянутую нуклеиновую кислоту, и его применение для лечения наследственной оптической нейропатии Лебера. Нуклеиновая кислота содержит последовательность нуклеотидов SEQ ID No. 1 и последовательность, кодирующую пептид митохондриальной локализации субъединицы 8 цитохром-с-оксидазы. Изобретения позволяют уменьшить уровень активных форм кислорода в клетке по меньшей мере на 25 % и уровень митохондриального кальция по меньшей мере на 29 %. Изобретения могут быть использованы для заместительной генной терапии наследственной оптической нейропатии Лебера. 3 н. и 8 з.п. ф-лы, 11 ил., 2 табл., 8 пр.
1. Нуклеиновая кислота для аллотопической экспрессии гена MT-ND4, содержащая последовательность нуклеотидов SEQ ID No. 1 и последовательность, кодирующую пептид митохондриальной локализации субъединицы 8 цитохром-с-оксидазы (белка Cox8).
2. Нуклеиновая кислота по п. 1, в которой пептид митохондриальной локализации белка Cox8 содержит последовательность SEQ ID No. 6.
3. Нуклеиновая кислота по п. 1 или 2, в которой последовательность SEQ ID No. 5 кодирует пептид митохондриальной локализации белка Cox8.
4. Экспрессионный вектор для экспрессии в эукариотических клетках, содержащий нуклеиновую кислоту по любому из пп. 1-3.
5. Вектор по п. 4, представляющий собой плазмидный экспрессионный вектор, содержащий в соответствии с физической и генетической картой, представленной на Фиг. 4, следующие элементы:
- участок начала репликации ori;
- левый инвертированный концевой повтор ITR;
- цитомегаловирусный промотор CMV promoter;
- последовательность интрона гена b-глобина человека;
- последовательность, кодирующую последовательность SEQ ID No. 6 пептида митохондриальной локализации (MTS) белка COX8;
- последовательность ND4opt SEQ ID No. 1;
- последовательность сигнала полиаденилирования poly(A);
- правый инвертированный концевой повтор ITR;
- промотор гена устойчивости к ампициллину AmpR promoter;
- ген устойчивости к антибиотику ампициллину AmpR.
6. Вектор по п. 5, содержащий последовательность SEQ ID No. 5 в качестве последовательности, кодирующей SEQ ID No. 6.
7. Вектор по любому из пп. 4-6, представляющий собой вирусный экспрессионный вектор.
8. Вектор по п. 7, представляющий собой аденоассоциированный вирус.
9. Вектор по п. 8, представляющий собой аденоассоциированный вирус 6 серотипа.
10. Вектор по п. 9, нарабатываемый в суспензионных клеточных культурах HEK293.
11. Применение вектора по любому из пп. 4-10 для лечения наследственной оптической нейропатии Лебера.
| US 11034954 B2, 15.06.2021 | |||
| US 2022259619 A1, 18.08.2022 | |||
| WO 2019241206 A1, 19.12.2019 | |||
| YU-WAI-MAN P | |||
| et al., Bilateral Visual Improvement with Unilateral Gene Therapy Injection for Leber Hereditary Optic Neuropathy | |||
| Science Translational Medicine, 09.12.2020 | |||
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| BONNET C | |||
| et al | |||
| The optimized allotopic expression | |||
Авторы
Даты
2023-12-06—Публикация
2023-10-17—Подача