ОБЛАСТЬ ТЕХНИКИ
Настоящее изобретение относится к биотехнологии. Описан рекомбинантный аденоассоциированный вектор rAAV на основе серотипа AAV.PHP.B, содержащий полинуклеотид, кодирующий белок выживаемости двигательных нейронов SMN (далее - AAV.PHP.B.SMN), предназначенный для модулирования двигательной функции у субъекта с нарушением двигательных нейронов, в частности для лечения спинальной мышечной атрофии.
УРОВЕНЬ ТЕХНИКИ
Спинальная мышечная атрофия - аутосомно-рецессивное наследственное нервно-мышечное заболевание, чаще проявляющееся у младенцев и детей, причиной которого является постепенное и необратимое нарушение функций двигательных нейронов в передних рогах спинного мозга, приводящее к симметричному ослабеванию и затем к атрофии мышц. Пациент со СМА постепенно теряет способность ползать, ходить, управлять собственным телом, самостоятельно сидеть, есть, глотать и дышать [1].
Генетической причиной СМА являются мутации или делеции гена SMN1, расположенного на длинном плече 5-й хромосомы (5q11.2-q13.3). Ген SMN1 кодирует белок выживания моторного нейрона SMN (survival of motor neuron), необходимый для работы двигательных нейронов. Примерно у 96% пациентов СМА вызвана гомозиготными делециями экзонов 7 и 8 гена SMN1 или, в большинстве случаев, экзона 7 [2].
Основным фактором, определяющим степень тяжести и прогноз пациентов со СМА, считается количество копий гена SMN2 (центромерная копия SMN1). Ген SMN2 тоже продуцирует полноразмерный функциональный белок SMN, но в относительно малых количествах по сравнению с геном SMN1 (до 10%). Чем больше копий гена SMN2, тем, как правило, менее выражены клинические симптомы СМА [3].
Таким образом, утрата гена SMN1 и, как следствие, сниженный уровень белка SMN частично компенсируются SMN-белком гена SMN2. Данный феномен объясняет фенотипическую вариабельность внутри СМА. В соответствии с пониманием компенсаторной роли гена SMN2 в современную клиническую классификацию СМА входит и число копий SMN2 (Таблица 1) [3].
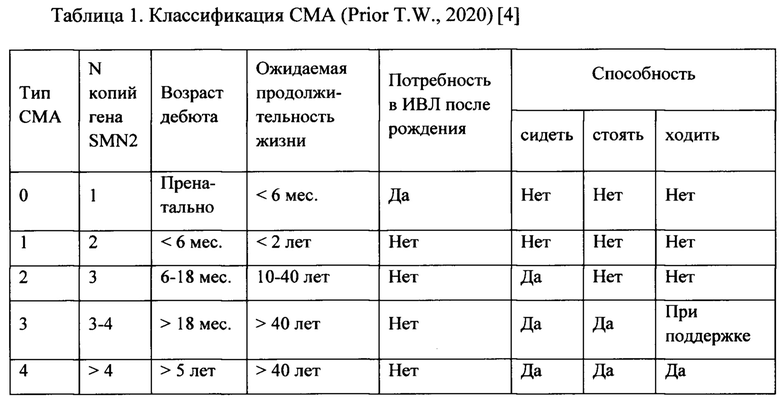
Наиболее неблагоприятные формы СМА с летальным исходом проявляются у младенцев и детей. Взрослые формы СМА (IV типа, возраст дебюта от 5 лет), отличаются более благоприятным клиническим течением. Двигательные нарушения у взрослых носят мягкий характер, больные не испытывают трудностей с дыханием и питанием, сохраняют способность ходить в зрелом возрасте.
СМА I типа (болезнь Верднига-Гоффманна) является одной из наиболее тяжелых и распространенных форм этого нейродегенеративного расстройства, на нее приходится более 60% больных СМА. Характеризуется ранним началом, в возрасте до 6 месяцев, и смертью от дыхательной недостаточности до достижения 2-летнего возраста. Дети, страдающие болезнью Верднига-Гоффманна, не способны держать голову, переворачиваться, сидеть без поддержки. Проксимальная симметричная мышечная слабость, отсутствие моторного развития и мышечная гипотония - основные клинические признаки СМА типа I. Наблюдаются трудности с глотанием и сосанием, что также способствует снижению защиты дыхательных путей и увеличивает риск развития аспирационной пневмонии.
Перспективным способом лечения СМА является генная терапия, нацеленная на доставку функционального гена SMN1 в ЦНС [5]. В 2019 году Управление по санитарному надзору за качеством пищевых продуктов и медикаментов США (FDA) одобрило первый лекарственный препарат для генной терапии СМА I степени у детей младше 2 лет ZOLGENSMA® (онасемноген абепарвовек) (группа компаний Новартис) [6]. ZOLGENSMA® является суспензией для внутривенного введения, содержащей рекомбинантный самокомплементарный аденоассоциированный вирусный вектор AAV9, способный преодолевать гематоэнцефалический барьер и проникать в клетки ЦНС, содержащий полинуклеотид, кодирующий белок SMN. Клинически доказано (STR1VE-US (NCT03306277)), что однократная инфузия препарата способна восстановить SMN-экспрессию в моторных нейронах, лишенных функционального гена SMN1. Структура экспрессионной кассеты лекарственного препарата ZOLGENSMA®: левый ITR, CMV-энхансер, промотор гена актина курицы (СВ-промотор), интрон позднего гена 16S субъединицы вируса SV40 (SV40), SMN1, сигнал полиаденилирования BGH, правый ITR.
Аденоассоциированные вирусные векторы (rAAV) считаются самыми безопасными и одними из наиболее широко используемых вирусных векторов для переноса генов in vivo. Они способны инфицировать клетки различных тканей, обеспечивая мощную и устойчивую экспрессию трансгена, являются непатогенными и и имеют низкий профиль иммуногенности [7-9].
Аденоассоциированный вирус (AAV) принадлежит к роду Dependoparvovirus семейства вирусов Parvoviridae. Представляет собой небольшой (20 нм), неспособный к самостоятельной репликации, безоболочечный вирус. У человека и приматов описано множество различных серотипов AAV. Известные серотипы могут инфицировать клетки различных видов тканей. Тканевая специфичность определяется серотипом белков капсида, поэтому векторы на основе аденоассоциированого вируса конструируют, задавая необходимый серотип.
Геном аденоассоциированного вируса содержит (+или -) одноцепочечную ДНК (ssDNA) длиной около 4,7 тысяч нуклеотидов. На концах молекулы геномной ДНК располагаются инвертированные концевые повторы (англ. inverted terminal repeats, ITRs). Геном содержит две открытые рамки считывания (англ. ORF): Rep и Сар, содержащие в себе несколько альтернативных рамок считывания, кодирующих различные белковые продукты. Продукты Rep имеют важное значение для репликации AAV, при этом ген Сар, помимо других альтернативных продуктов, кодирует 3 капсидных белка (VP1, VP2 и VP3). Белки VP1, VP2 и VP3 находятся в соотношении 1:1:10, образуя икосаэдрический капсид [10]. При образовании рекомбинантного вектора AAV (rAAV) кассета экспрессии, фланкированная ITR, упаковывается в капсид AAV. Гены, необходимые для репликации AAV, не входят в кассету.
Эффективности опосредованного rAAV переноса генов препятствует необходимость конвертации одноцепочечного (оцДНК) генома в двухцепочечную ДНК (дцДНК) перед экспрессией. Стадию конвертации оцДНК в дцДНК можно исключить посредством использования векторов, содержищих самокомплементарный геном (scAAV), кодирующая область которых была разработана для формирования внутримолекулярной двухцепочечной матрицы ДНК. При scAAV-инфицировании вместо клеточно-опосредованного синтеза второй цепи ДНК или спаривания цепей двух вирусных частиц, две комплементарные половины scAAV будут связываться с образованием одной двухцепочечной единицы ДНК, готовой к экспрессии. Недостаток этой конструкции заключается в том, что вместо полной кодирующей емкости rAAV (4,7-6 кб) scAAV может содержать только около половины этой емкости (≈2,4 кб) [11]. Еще одним важным недостатком является повышенная по сравнению с ssAAV иммуногенность: scAAV индуцируют более выраженный иммунный ответ путем передачи сигналов через TLR9 [12].
Из уровня техники известны как scAAV векторы (WO 2010071832, RU 2743398, RU 2603740, CN 112725344), так и ssAAV векторы (ЕА 201992032, WO 2019011817, WO 2020127813, WO 2021246909) для доставки гена SMN1 в ЦНС субъекта, страдающего спинальной мышечной атрофией.
Анализ уровня техники показал, что одной из насущных проблем в лечении спинальной мышечной атрофии генно-терапевтическими препарами являются иммуннотоксические реакции. Наиболее частым проявлением таких реакций считается гепатотоксичность, которая характеризуется значительным повышением активности печеночных трансаминаз. Гепатотоксичность наблюдается почти у трети пациентов, получивших геннотерапевтический препарат для лечения СМА, у некоторых из них введение вирусного вектора приводит к тяжелому повреждению печени с развитием острой печеночной недостаточности [13]. Нежелательные иммунотоксические реакции связаны с высокой дозой вводимых вирусных векторов. Иммунотоксические реакции развиваются несмотря на профилактическое введение глюкокортикостероидов. В исследованиях, оценивающих эффективность и безопасность разных доз геннотерапевтических препаратов, наблюдается коррелляция между дозой и иммунным ответом на препарат [14, 15]. Учитывая дозозависимый харакатер иммунотоксических реакций, снижение дозы геннотерапевтического препарата для лечения СМА позволит снизить частоту нежелательных реакций у пациентов. Одним из способов снижения дозы является оптимизация векторов для увеличения трансдукции в целевой популяции клеток.
Цель настоящего изобретения - оптимизация вектора для доставки гена SMN1, используемого в геннотерапевтическом препарате ZOLGENSMA®, который является наиболее близким аналогом настоящего изобретения, путем замены капсида AAV9 на капсид AAV.PHP.B. Еще одной целью настоящего изобретения явилась оптимизация вышеупомянутого вектора путем замены как капсида AAV, так и кассеты экспрессии с одновременной заменой самокомплементарного генома (sc) на одноцепочесный геном (ss).
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Настоящее изобретение основано на открытии того, что рекомбинантный аденоассоциированный вектор AAV.SMN на основе серотипа AAV.PHP.B (далее -AAV.PHP.B.SMN) способствует более эффективной терапии СМА по сравнению с вектором AAV9.SMN. Рекомбинантный аденоассоциированный вирус AAV.PHP.B (SEQ ID NO 1), отличается от AAV9 (SEQ ID NO 2) инсерцией гептапептидной аминокислотной вставки TLAVPFK между аминокислотами 588 и 589 VP1 капсида AAV9, что обеспечивает повышение проницаемости гематоэнцефалического барьера и селективную трансдукцию клеток ЦНС в сравнении с AAV9 [16]. Из уровня техники неизвестны рекомбинантные аденоассоциированные векторы AAV на основе серотипа AAV.PHP.B, способные доставлять ген SMN1 в ЦНС.
Проведенное нами исследование эффективности векторов AAV.PHP.B.SMN (пример 7) в сравнении с AAV.9.SMN в лечении СМА у мышей, лишенных гена SMN1, показало, что использование векторов AAV.PHP.B способствует большей выживаемости животных.
Настоящее изобретение относится к рекомбинантному аденоассоциированному вирусному (rAAV) вектору на основе серотипа AAV.PHP.B, содержащему полинуклеотид, кодирующий белок выживаемости двигательных нейронов (SMN).
В предпочтительном варианте осуществления изобретения белок SMN кодирован геном SMN1 (SEQ ID NO: 3). В другом предпочтительном варианте осуществления изобретения белок SMN может быть кодирован кодон-оптимизированным геном SMN1.
В предпочтительном варианте осуществления изобретения белок SMN содержит аминокислотную последовательность SEQ ID NO: 4.
В дополнительном варианте осуществления изобретения белок SMN содержит аминокислотную последовательность, которая обладает по меньшей мере 90% идентичностью с последовательностью SEQ ID NO: 4.
В предпочтительном варианте осуществления изобретение относится к вектору AAV.PHP.B.SMN, капсид которого включает белок VP1, имеющий аминокислотную последовательность SEQ ID NO: 1.
В дополнительных вариантах осуществления изобретение относится к вектору AAV.PHP.B.SMN, капсид которого включает белок VP1, имеющий аминокислотную последовательность SEQ ID NO: 1 с одной или несколькими точечными мутациями.
В одном из вариантов осуществления изобретение относится к вектору AAV.PHP.B.SMN, имеющему одноцепочечный геном, кодирующий белок SMN (ssAAV).
В предпочтительном варианте осуществления изобретение относится к вектору AAV.PHP.B.SMN, имеющему одноцепочечный геном (ssAAV), кодирующий белок SMN, причем указанный геном упакован в капсид, включающий белок VP1 AAV.PHP.B, имеющий аминокислотную последовательность SEQ ID NO: 1 или аминокислотную последовательность с одной или несколькими точечными мутациями, а экспрессионная кассета включает следующие элементы в направлении от 5'-конца к 3'-концу:
ITR;
CMV энхансер;
CMV промотер SMN1;
сигнал полиаденилирования hGH1;
ITR.
В еще более предпочтительном варианте осуществления изобретение относится к вектору AAV.PHP.B.SMN, имеющему одноцепочечный геном (ssAAV), кодирующий белок SMN, при чем указанный геном упакован в капсид, включающий белок VP1 AAV.PHP.B, имеющий аминокислотную последовательность SEQ ID NO: 1 или аминокислотную последовательность с одной или несколькими точечными мутациями, а экспрессионная кассета имеет последовательность SEQ ID NO: 5 или на 90% идентичную SEQ ID NO: 5.
В одном из вариантов осуществления изобретение относится к вектору AAV.PHP.B.SMN, имеющему самокомплементарный геном (scAAV), кодирующий SMN.
В предпочтительном варианте осуществления изобретение относится к вектору AAV.PHP.B.SMN, имеющему самокомплементарный геном (scAAV), кодирующий SMN, при чем указанный геном упакован в капсид, включающий белок VP1 AAV.PHP.B, имеющий аминокислотную последовательность SEQ ID NO: 1 или аминокислотную последовательность с одной или несколькими точечными мутациями, а экспрессионная кассета включает следующие элементы в направлении от 5'-конца к 3'-концу:
ITR
CMV энхансер;
СВ промотор;
SV40 интрон SMN1;
сигнал полиаденилирования BGH;
ITR.
В еще более предпочтительном варианте осуществления изобретение относится к вектору AAV.PHP.B.SMN, имеющему самокомплементарный геном (scAAV), кодирующий белок SMN, при чем указанный геном упакован в капсид, включающий белок VP1 AAV.PHP.B, имеющий аминокислотную последовательность SEQ ID NO: 1 или аминокислотную последовательность с одной или несколькими точечными мутациями, а экспрессионная кассета имеет последовательность SEQ ID NO: 6 или на 90% идентичную SEQ ID NO: 6.
В одном из вариантов осуществления изобретение относится к фармацевтической композиции, предназначенной для доставки гена SMN1 (SEQ ID NO: 3) в целевые клетки, содержащей вектор AAV.PHP.B.SMN, описанный выше, в сочетании с одним или несколькими фармацевтически приемлемыми эксципиентами.
В дополнительном варианте осуществления изобретение относится к применению вектора AAV.PHP.B.SMN или фармацевтической композиции, содержащей вектор AAV.PHP.B.SMN, для доставки гена SMN1 (SEQ ID NO: 3) в целевые клетки.
ТЕРМИНЫ И ОПРЕДЕЛЕНИЯ
Используемый в настоящем документе термин капсид AAV.PHP.B представляет собой самоорганизующийся капсид AAV, состоящий из трех белков: VP1, VP2, VP3. Белки капсида AAV.PHP.B транслируются из двух мРНК, формирующихся в результате альтернативного сплайсинга гена cap, который имеет нуклеотидную последовательность SEQ ID NO: 7, или последовательность, соответствующей ей по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 97%, по меньшей мере на 99%, которая кодирует аминокислотную последовательность VP1 SEQ ID NO: 1.
В некоторых вариантах осуществления изобретения «капсид AAV.PHP.B» включает в себя AAV, имеющий аминокислотную последовательность белков VP1 SEQ ID NO: 1, VP2 SEQ ID NO: 21 и VP3 SEQ ID NO: 22 с одной или несколькими точечными мутациями. Варианты точечных мутаций в последовательности белков капсида VP1-VP3 AAV.PHP.B представляет собой замену, по меньшей мере, одного аминокислотного остатка в белках VP1-VP3 на другой аминокислотный остаток.
Под «несколькими точечными мутациями» подразумеваются две, три, четыре, пять, шесть, семь, восемь, девять или десять точечных замен.
Дополнительные варианты осуществления изобретения включают замены (мутации), которые являются консервативными по природе, т.е. замены на аминокислоты, обладающие схожими физико-химическими свойствами боковых цепей. В частности, аминокислоты обычно делят на пять групп: (1) нейтральные: глицин, аланин, валин, лейцин, изолейцин, пролин; (2) кислые: глутамат и аспартат; (3) основные: лизин, аргинин, гистидин; (4) полярные: метионин, аспарагин, глутамин, цистеин, серии треонин; (5) ароматические: фенилаланин, тирозин, триптофан (таблица 2).

Например, достаточно обосновано предсказание о том, что выделенная замена лейцина на изолейцин или валин, аспартата на глутамат, треонина на серии или схожая консервативная замена аминокислоты на структурно родственную аминокислоту не окажет существенного влияния на биологическую активность. Например, полипептид, представляющий интерес, может включать вплоть до приблизительно 5-10 консервативных или неконсервативных аминокислотных замен, при условии, что желаемая функция молекулы остается незатронутой.
«Рекомбинантный AAV» или «rAAV» представляет собой устойчивую к ДНКазе вирусную частицу, содержащую два элемента, капсид AAV и векторный геном, содержащий по меньшей мере не кодирующие AAV последовательности, упакованные в капсид AAV. В некоторых вариантах осуществления изобретения капсид содержит около 60 белков, состоящих из белков VP1, белков VP2 и белков VP3, которые самоорганизуются с образованием капсида. Если не указано иное, «рекомбинантный AAV» или «rAAV» могут использоваться взаимозаменяемо в словосочетании «вектор rAAV». rAAV представляет собой «вирус, дефектный по репликации» или «вирусный вектор», так как в нем отсутствует какой-либо функциональный ген rep AAV или функциональный ген cap AAV, вследствие чего он не способен к репликации. В некоторых вариантах осуществления изобретения единственными последовательностями AAV являются последовательности инвертированных концевых повторов (ITR) AAV, обычно расположенные на крайних 5' и 3' концах векторного генома, чтобы обеспечить упаковку гена и регуляторных последовательностей, расположенных между ITR, внутрь капсида AAV.
Термин «устойчивый к нуклеазе» указывает на то, что капсид AAV собран вокруг экспрессионной кассеты, которая предназначена для доставки гена в клетку-хозяина, и защищает эти упакованные геномные последовательности от деградации (расщепления) во время этапов инкубации нуклеазы, предназначенных для удаления загрязняющих нуклеиновых кислот, которые могут присутствовать в процессе производства.
В определенных вариантах осуществления изобретения невирусные генетические элементы, используемые при продуцировании rAAV, будут упоминаться как плазмиды. Такие плазмиды представляют собой нуклеиновые кислоты, которые могут кодировать последовательности, необходимые для сборки rAAV, например белки капсида AAV, rep-белки, или вспомогательные белки необходимые для продуцирования rAAV, которые не упакованы в rAAV. Альтернативно, такая плазмида продукции может нести векторный геном, который упакован в rAAV.
Используемый в данном документе, термин «векторный геном» относится к последовательности нуклеиновой кислоты, упакованной внутрь капсида rAAV, который образует вирусную частицу. Такая последовательность нуклеиновой кислоты содержит последовательности инвертированных концевых повторов (ITR) AAV. В приведенных в данном документе примерах векторный геном содержит, как минимум, от 5' до 3', 5' ITR AAV, кодирующую последовательность (-и) и 3' ITR AAV. Могут быть выбраны ITR из AAV2, источника AAV, отличного от капсида, или отличные от полноразмерных ITR. В некоторых вариантах осуществления изобретения ITR получены из того же источника AAV, что и AAV, который обеспечивает функцию гер (репликации) во время продуцирования или транскомплементации AAV. Кроме того, могут использоваться другие ITR. Кроме того, векторный геном содержит регуляторные последовательности, которые направляют экспрессию генных продуктов. Подходящие компоненты векторного генома более подробно описаны ниже.
В некоторых вариантах осуществления изобретения термин «экспрессионная кассета» относится к молекуле нуклеиновой кислоты, кодирующей последовательность белка SMN и последовательности контроля экспрессии, которые направляют экспрессию последовательностей SMN в клетке-хозяине (например, промотор, энхансер, полиА), при этом кассета может быть упакована в капсид вирусного вектора (например, вирусную частицу). Такая экспрессионная кассета, предназначенная для создания вирусного вектора, содержит последовательности SMN, описанные в настоящем изобретении, фланкированные сигналами упаковки вирусного генома и другими последовательностями контроля экспрессии, описанными ниже. Например, для вирусного вектора AAV сигналами упаковки являются 5'-инвертированный концевой повтор (ITR) и 3'-ITR. В некоторых вариантах осуществления изобретения термин «трансген» можно использовать взаимозаменяемо с «экспрессионной кассетой».
Используемый в настоящем документе термин «SMN» включает в себя любую изоформу SMN, которая восстанавливает желаемую функцию, уменьшает симптом или обеспечивает другой желаемый физиологический результат при доставке композиции или в способе, представленных в настоящем документе. В приведенных в данном документе примерах используется самая длинная изоформа, изоформа D, которая, как полагают, является преобладающим транскриптом, продуцируемым геном, у пациента, не затронутого дефицитом или дефектом SMN. Изоформа D представляет белок из 294 аминокислот [см., например, регистрационный номер NCBI NM 000344.4; NP 000335; ENSEMBL ID ENST000003 80707], последовательность белка воспроизведена в SEQ ID NO: 4, и кодирующая последовательность воспроизведена в SEQ ID NO: 3. Однако может быть выбрана другая изоформа белка SMN.
В настоящем документе также представлены вирусные векторы rAAV.PHP.B.SMN, состоящие из внешнего капсида AAV.PHP.B и внутреннего генома ДНК, кодирующего белок SMN. В одном из вариантов внутри капсида упакован геном одноцепочечной ДНК (оцДНК). В другом из вариантов внутри капсида упакован самокомплементарный геном ДНК (дцДНК). Геном оцДНК и дцДНК состоит из трансгена выживания моторного нейрона (SMN) человека, фланкированного двумя инвертированными концевыми повторами (ITR) AAV. Трансген SMN включает энхансер, промотор, интрон, кодирующую последовательность гена SMN1 и сигнал полиаденилирования (полиА сигнал). ITR являются генетическими элементами, ответственными за репликацию и упаковку генома во время продуцирования вектора, и являются единственными вирусными цис-элементами, необходимыми для генерации rAAV. В геноме оцДНК экспрессия кодирующей последовательности SMN управляется промотором/энхансером, полученным из немедленной ранней (IE) области цитомегаловируса (hCMV) человека. Транскрипция с этого промотора усиливается присутствием интрона гена hBG1 (ген субъединицы гемоглобина гамма-1). ПолиА сигнал hGH1 (сигнал полиаденилирования гена гормона роста человека) включается для обеспечения терминации транскриптов мРНК SMN человека. В самокомплементарном геноме экспрессия кодирующей последовательности SMN управляется промотором/энхансером, полученным из гена бета-актина курицы и немедленной ранней (IE) области цитомегаловируса (hCMV) человека. Транскрипция с этого промотора/энхансера усиливается присутствием интрона позднего гена 16S субъединицы вируса SV40. ПолиА сигнал BGH (сигнал полиаденилирования бычьего гормона роста) включается для обеспечения терминации транскриптов мРНК SMN человека.
Под «функциональным SMN1» или «SMN1» подразумевается ген, который кодирует белок SMN, который обеспечивает по меньшей мере около 50%, по меньшей мере около 75%, по меньшей мере около 80%, по меньшей мере около 90%, или около 100%, или более чем 100% от уровня биологической активности нативного белка выживания моторных нейронов. Кроме этого, ген SMN2, центромерная копия SMN1, также кодирует белок SMN, но в меньших количествах по сравнению с SMN1. На основании количества копий SMN2 пациенты, у которых отсутствует функциональный ген SMN1, проявляют фенотипическую вариабельность СМА в различной степени. Таким образом, в некоторых случаях может быть желательным, чтобы белок SMN обеспечивал менее чем 100% биологической активности нативного белка SMN.
В одном варианте осуществления изобретения аминокислотная последовательность функционального SMN представляет собой последовательность SEQ ID NO: 4 или последовательность, имеющую по меньшей мере 90% идентичности с ней.
Термин «процент (%) идентичности», «идентичность последовательности», «процент идентичности последовательности» или «на … процентов идентичная» в контексте последовательностей нуклеиновых кислот относится к остаткам в двух последовательностях, которые являются аналогичными при выравнивании на совпадение. Сравнение идентичности последовательностей может выполняться по всей длине генома, желательна полная длина последовательности, кодирующей ген, или фрагмента по меньшей мере около от 500 до 5000 нуклеотидов. Однако также может быть необходима идентичность между более мелкими фрагментами, например, из по меньшей мере около девяти нуклеотидов, как правило, из по меньшей мере около от 20 до 24 нуклеотидов, из по меньшей мере около от 28 до 32 нуклеотидов, из по меньшей мере около 36 или более нуклеотидов.
Процент идентичности для аминокислотных последовательностей определяется по всей длине белка, полипептида, от около 32 аминокислот до около 330 аминокислот, или для их пептидного фрагмента, или кодирующих последовательностей соответствующих последовательностей нуклеиновых кислот. Длина подходящего аминокислотного фрагмента может составлять по меньшей мере 8 аминокислот и может быть до около 700 аминокислот. Как правило, когда речь идет об «идентичности», «гомологии» или «сходстве» между двумя различными последовательностями, «идентичность», «гомология» или «сходство» определяются по отношению к «выровненным» последовательностям. «Выровненные» последовательности или «выравнивания» относятся к множеству нуклеотидных последовательностей или белковых (аминокислотных) последовательностей, часто содержащих коррекции для отсутствующих или дополнительных оснований или аминокислот по сравнению с эталонной последовательностью. Идентичность аминокислотных последовательностей может быть определена путем выравнивания последовательностей с использованием различных алгоритмов и/или компьютерных программ, известных в данной области техники или коммерчески доступных (например, BLAST, ExPASy; ClustalO; FASTA; используя, например, алгоритм Нидлмана-Вунша, алгоритм Смита-Уотермана). Многочисленные программы выравнивания последовательностей также доступны и для нуклеотидных последовательностей. К примерам таких программ относится Clustal Omega, Clustal W, CAP Sequence Assembly, BLAST, MAP и MEME.
Вектор AAV содержит последовательности ITR AAV. В одном варианте осуществления изобретения ITR взяты из AAV, отличного от того, который предоставляет капсид. В предпочтительном варианте осуществления изобретения (в целях ускорения регистрации лекарственного препарата) могут быть использованы последовательности ITR из AAV дикого типа или модифицированные последовательности ITR. В качестве ITR дикого типа может быть выбран ITR из AAV2. Если ITR из AAV2, а капсид AAV - из другого источника AAV, результирующий вектор может быть назван псевдотипированным. Как правило, векторный геном AAV содержит 5' ITR AAV, кодирующие последовательности SMN, регуляторные последовательности и 3' ITR AAV. Однако и другие конфигурации этих элементов могут быть подходящими. Модифицированнные последовательности ITR могут быть получены путем внесения мутаций в ITR, полученные из AAV дикого типа. Модифицированные последовательности ITR могут способствовать формированию дцДНК.
Экспрессионная кассета, как правило, в качестве последовательности контроля экспрессии содержит промоторную последовательность, расположенную, например, между выбранной 5' ITR последовательностью и кодирующей последовательностью SMN. В описанном в настоящем документе геноме оцДНК экспрессия кодирующей последовательности SMN управляется промотором/энхансером, полученным из немедленной ранней (IE) области цитомегаловируса (hCMV) человека. В описанном в настоящем документе самокомплементарном геноме экспрессия кодирующей последовательности SMN управляется промотором/энхансером, полученным из гена бета-актина курицы и немедленной ранней (IE) области цитомегаловируса (hCMV) человека.
Термин «промотор», используемый в настоящем документе, в частности, относится к элементу ДНК, который способствует транскрипции полинуклеотида, с которым функционально связан промотор. Промотор может также составлять часть элемента «промотор/энхансер». Хотя физические границы между элементами «промотор» и «энхансер» не всегда ясны, термин «промотор» обычно относится к месту на молекуле нуклеиновой кислоты, с которым связывается РНК-полимераза и/или связанные с ней факторы, и с которого инициируется транскрипция. Энхансеры усиливают активность промотора во времени, а также пространственно. В данной области известно множество промоторов, которые транскрипционно активны в широком диапазоне типов клеток. Промоторы могут быть разделены на два класса: на функционирующие конститутивно и регулируемые индукцией или снятием репрессии. Для экспрессии белка пригодны оба класса. Промоторы, которые используются для продукции высокого уровня полипептидов в эукариотических клетках и, в частности, в клетках млекопитающих, должны быть сильными и, предпочтительно, должны быть активными в широком диапазоне типов клеток. Сильные конститутивные промоторы, которые способны запускать экспрессию во многих типах клеток, хорошо известны в данной области и, поэтому, нет необходимости в их подробном описании в данном документе. В соответствии с одним из вариантов настоящего изобретения предпочтительно использовать промотор цитомегаловируса (CMV). Промотор или промотор/энхансер, полученные из немедленной ранней (IE) области цитомегаловируса (hCMV) человека, в особенности подходят в качестве промотора в экспрессионной кассете по настоящему изобретению. Немедленная ранняя (IE) область цитомегаловируса (hCMV) человека и полученные из нее функциональные запускающие экспрессию фрагменты и/или функциональные усиливающие экспрессию фрагменты, например, описаны в ЕР 0173177 и ЕР 0323997, а также хорошо известны в данной области. В соответствии с другим вариантом настоящего изобретения предпочтительно использовать промотор/энхансер, полученный из гена бета-актина курицы и немедленной ранней (IE) области цитомегаловируса (hCMV) человека.
В дополнение к промотору/энхансеру экспрессионная кассета и/или вектор могут содержать одну или более других подходящих последовательностей инициации, терминации транскрипции, сигналов эффективного процессирования РНК, такие как сигналы сплайсинга и полиаденилирования (полиА); последовательности, которые стабилизируют цитоплазматическую мРНК, например WPRE; последовательности, которые повышают эффективность трансляции (т.е. консенсусная последовательность Козак); последовательности, которые повышают стабильность белка; и, при желании, последовательности, которые усиливают секрецию кодируемого продукта. Характер таких контролирующих последовательностей различается в зависимости от организма-хозяина; в прокариотах такие контролирующие последовательности, как правило, включают промотор, сайт связывания рибосомы, а также последовательности терминации транскрипции; в эукариотах, как правило, такие контролирующие последовательности включают промоторы и последовательности терминации транскрипции. Термин «контролирующие последовательности» включает, как минимум, все компоненты, наличие которых имеет важное значение для экспрессии и процессинга, и может также включать дополнительные компоненты, присутствие которых является полезным, например, лидирующие последовательности и последовательности слившихся клеток.
В одном из аспектов настоящее изобретение относится к фармацевтической композиции для доставки гена SMN1 в целевые клетки, которая включает вышеуказанный рекомбинантный вирус на основе AAV.PHP.B в сочетании с одним или несколькими фармацевтически приемлемыми эксципиентами.
Термин «фармацевтическая композиция» обозначает композицию, включающую в себя вышеуказанный рекомбинантный вирус на основе AAV.PHP.B по изобретению и, по крайней мере, один из компонентов, выбранных из группы, состоящей из фармацевтически приемлемых и фармакологически совместимых эксципиентов, таких как наполнители, растворители, разбавители, носители, средства доставки, консерванты, стабилизаторы, эмульгаторы, суспендирующие агенты, загустители, регуляторы пролонгированной доставки, выбор и соотношение которых зависит от способа назначения и дозировки. Носитель для инъекций обычно является жидким. Носитель для других способов введения может быть твердым или жидким. В качестве инъекционной среды предпочтительно использовать воду, содержащую добавки, общепринятые для инъекционных растворов, такие как буферные и изотонические агенты, стабилизаторы и солюбилизаторы. Фармацевтические композиции по настоящему изобретению и способы их изготовления являются очевидными для специалистов в этой области. Производство фармацевтических композиций предпочтительно должно соответствовать требованиям GMP (надлежащей производственной практики).
В одном из аспектов настоящее изобретение относится к применению вышеуказанного рекомбинантного вируса на основе AAV.PHP.B или вышеуказанной композиции для доставки гена SMN1 в целевые клетки. Любой способ введения рекомбинантного вируса на основе AAV, принятый в данной области, может соответствующим образом использоваться для вышеуказанного рекомбинантного вируса на основе AAV.PHP.B по настоящему изобретению.
Рекомбинантный вирус на основе AAV.PHP.B по изобретению предпочтительно вводят в клетку в биологически эффективном количестве. «Биологически эффективное» количество рекомбинантного вируса представляет собой количество, которое достаточно, чтобы вызвать инфекцию (или трансдукцию) и экспрессию гетерологичной последовательности нуклеиновой кислоты в клетке. Если вирус вводят в клетку in vivo (например, вирус вводят субъекту, как описано ниже), «биологически эффективное» количество вирусного вектора представляет собой количество, которое достаточно, чтобы вызвать трансдукцию и экспрессию гетерологичной последовательности нуклеиновой кислоты в клетке-мишени. Вышеуказанный рекомбинантный вирус на основе AAV.PHP.B не используется для модификации генетической целостности клеток зародышевой линии человека.
КРАТКОЕ ОПИСАНИЕ ФИГУР
На фиг. 1А-1С приведены результаты определения экспрессии SMN1 на уровне мРНК после трансфекции плазмидами sc-AAV-SMN1 и ss-AAV-SMN1.
На фиг. 1А представлены результаты определения количества РНК SMN1 в реакционной смеси.
На фиг. 1В приведены результаты определения количества РНК hGAPDH в реакционной смеси.
На фиг. 1С показана нормализация уровня РНК SMN1 к hGAPDH с учетом фоновой экспрессии в контрольных клетках без добавления трансфецирующего агента и плазмид (КО).
На фиг. 2А и 2В приведены результаты определения белка SMN после трансфекции плазмидами sc-AAV-SMN1 и ss-AAV-SMN1.
На фиг. 2А приведены результаты определения белка SMN методом Вестерн-блоттинга:
1 - Блот после инкубации с антителами Anti-SMN/Gemin 1. М - белковый маркер PageRuler Plus Prestained Protein Ladder; для каждого варианта трансфекции sc-AAV-SMN1, ss-AAV-SMN1 и контроля без добавления плазмиды (КО) загружено по 2 клеточного лизата, приготовленных независимо.
2 - Блот после инкубации с антителами, узнающими белок домашнего хозйства β-Actin (8H10D10) Mouse mAb.
На фиг. 2В приведены результаты определения белка SMN в образцах методом ИФА.
При изучении трансляции целевого белка SMN методом ИФА проведено не менее трех независимых экспериментов по трансфекции, включая контроль без добавления трансфецирующего агента и плазмид, определены среднее значение, стандартное отклонение (SD) и коэффициент вариации (CV);
sc-AAV-SMN1 - клетки НЕК293 после трансфекции плазмидой sc-AAV-SMN1;
ss-AAV-SMN1- клетки НЕК293 после трансфекции плазмидой ss-AAV-SMN1;
КО - клетки НЕК293 без добавления плазмид и трансфецирующего агента.
ОСУЩЕСТВЛЕНИЕ ИЗОБРЕТЕНИЯ
Материалы и методы
Методы молекулярного клонирования
Некоторые элементы генетических конструкций, включая последовательность гена SMN1, получены методом химического синтеза в компании Atum Dna2.0.
Для манипуляций с ДНК используют стандартные методы [17]. Для выделения плазмидной ДНК клетки Е. coli растят на селективной среде с добавлением антибиотика. Плазмидную ДНК выделяют с помощью коммерческих наборов. Необходимые фрагменты ДНК получают с помощью ПЦР или эндонуклеаз рестрикции. Электрофоретическое разделение фрагментов ДНК проводят в агарозном геле. Фрагменты ДНК нужного размера выделяют из геля коммерческими наборами. Концентрацию ДНК определяют методом спектрофотометрии. После лигирования фрагментов ДНК проводят электропорацию бактериальных клеток. Трансформированные клетки отбирают на селективной среде с добавлением антибиотика, затем из них выделяют плазмидную ДНК. Корректность генетических конструкций подтверждена рестрикционным картированием, ПЦР и секвенированием по Сэнгеру. Анализ последовательностей ДНК проведен в программе SnapGene 3.2.1.
Культивирование клеток НЕК293
Разморозка клеток Нек293
Клетки НЕК293 быстро, в течение 40-60 сек, размораживают на водяной бане при температуре 37°С. Откручивают крышку, отбирают весь объем клеточной суспензии и переносят в 15 мл пробирку типа Falcon в 8 мл среды ЕМЕМ с глутамином с добавлением 10% FBS. Аккуратно пипетируют, после чего центрифугируют в течение 5 мин со скоростью 170g при комнатной температуре (далее - RT). Отбирают супернатант, оставляют в пробирке 2 мл среды, осадок тщательно пипетируют. Оценивают концентрацию и жизнеспособность клеток прямым подсчетом в камере Горяева.
Клетки рассаживают в культуральные флаконы Т75 (площадь 75 см2) с вентилируемой крышкой в концентрации 3-5×104 кл/мл в 15 мл ЕМЕМ с 10% FBS без антибиотиков. Клетки культивируют в СО2-инкубаторе при температуре 37°С во влажной атмосфере 5% СО2 в течение 48-72 часов.
Пассирование Hek293
Пассирование клеток проводят по достижении конфлюэнтности во флаконе 80-90%. Из флаконов Т75 удаляют старую культуральную среду. Клетки промывают 10 мл DPBS, после чего в каждый флакон вносят по 3 мл 0,25% раствора трипсина-0,53 мМ ЭДТА. Смачивают поверхность флакона покачиванием и инкубируют 10 мин в СО2-инкубаторе при температуре 37°С. Полноту открепления клеток контролируют под микроскопом. Вносят в каждый флакон по 10 мл полной ростовой среды для инактивации трипсина, аккуратно промывают дно флакона и переносят клетки в 50 мл флаконы типа Falcon. Клетки тщательно ресуспендируют и центрифугируют в течение 5-10 мин со скоростью 170-300g при RT. Отбирают супернатант, оставляют во флаконах по 5-10 мл клеточной суспензии, пипетируют, измеряют концентрацию и оценивают жизнеспособность на камере Горяева. Клетки рассаживают во флаконы Т75 в концентрации 3,0-5,0×104 кл/мл. Необходимое количество клеток переносят в 15 мл полной ростовой среды и культивируют в СО2-инкубаторе в течение 48-72 часов при температуре 37°С в атмосфере 5% СО2.
Трансфекция клеток НЕК293
Трансфекцию TurboFect Transfection Reagent (Sigma-Aldrich) (далее - TurboFect) проводят согласно рекомендациям производителя. Для трансфекции клетки рассеивают в лунки 6-луночного планшета в концентрации 1×105 клеток/мл в 3 мл полной ростовой среды ЕМЕМ. Инкубируют 24-48 ч в СО2-инкубаторе при температуре 37°С в атмосфере 5% СО2. Трансфекцию проводят при плотности монослоя 70-90%. Для приготовления трансфецирующей смеси в 2 отдельные микропробирки типа Eppendorf вносят по 150 мкл среды ЕМЕМ, в одну добавляют 5 мкл TurboFect, в другую вносят 1,5 мкг плазмидной ДНК, перемешивают на вортексе 5 сек. Затем готовят ДНК-липидный комплекс, смешав 1:1 смесь ДНК и разбавленный реагент Turbofect, перемешивают на вортексе 5 сек и инкубируют 20 мин при RT. Трансфекционную смесь вносят в соответствующие лунки культурального планшета и инкубируют в течение 48 часов в СО2-инкубаторе при температуре 37°С в атмосфере 5% СО2. Отрицательным контролем служат клетки, нетрансфицированные плазмидной ДНК.
Для каждой экспрессионной кассеты проводят не менее трех независимых повторов трансфекции в одинаковых условиях.
Отбор клеток НЕК293, приготовление лизатов, выделения РНК и синтез кДНК По окончанию культивирования клеток ростовую среду отбирают и утилизируют, клетки аккуратно промывают 2 мл раствора DPBS. После удаления DPBS в лунки вносят по 1 мл раствора трипсина-ЭДТА 0,25%, инкубируют 10 мин при 37°С, добавляют 2 мл DPBS, аккуратно перемешивают и переносят суспензию клеток в 15 мл пробирку, добавляют 5 мл DPBS, перемешивают, центрифугируют 5 мин на скорости 250 g при 4°С, удаляют супернатант. Клетки промывают 5 мл DPBS, центрифугируют 5 мин на скорости 250 g при 4°С, удаляют супернатант. Затем клетки ресуспендируют в 2,1 мл DPBS, переносят по 1 мл в 1,5 мл микропробирки, центрифугируют 5 мин на скорости 250 g при 4°С, аккуратно удаляют супернатант. Далее клетки используют для выделения РНК/приготовления лизатов или замораживают (-20°С).
Непосредственно перед началом работы готовят лизирующий буфер RIPA. В пробирки с клетками добавляют лизирующий буфер, инкубируют 10 минут на льду. Затем лизаты центрифугируют 20 минут при +4°С, 18000 g, аликвотируют по 20 мкл и переносят в низкотемпературный морозильник (-80°С).
Тотальную РНК выделяют с помощью набора Thermo Scientific GeneJET RNA Purification Kit согласно инструкции производителя. Синтез кДНК проводят с помощью набора QuantiTect Reverse Transcription Kit согласно инструкции производителя.
Анализ экспрессии гена SMN1
ОТ-ПЦР в реальном времени
Полимеразную цепную реакцию проводят на амплификаторе Real-Time CFX96 Touch (BioRad). Для постановки рвПЦР на основе интеркалирующего красителя SYBR используют готовую смесь для ПЦР SsoAdvancedTM Universal SYBR® Green Supermix. Для построения калибровочных кривых в реакцию общим объемом 10 мкл вносят 5 мкл SsoAdvanced universal SYBR Green supermix, по 0,5 мкл прямого и обратного праймера (10 мкМ), 2 мкл деионизованной воды, 2,5 мкл стандартов плазмидной ДНК. Для анализа образцов кДНК в реакцию общим объемом 10 мкл вносят 5 мкл SsoAdvanced universal SYBR Green supermix, по 0,5 мкл прямого и обратного праймера (10 мкМ), 3,5 мкл деионизованной воды, 1 мкл образцов кДНК. Результаты амплификации оценивают при помощи программы Bio-Rad CFX Manager. С использованием соответствующих стандартных кривых рассчитывают содержание кДНК SMN1 и hGAPDH в образцах клеток НЕК293 после трансфекции (количество молекул кДНК/1 мкл реакционной смеси).
Для подсчета уровня экспрессии гена SMN1 (L) концентрацию кДНК SMN1 нормализуют к концентрации гена домашнего хозяйства hGAPDH:
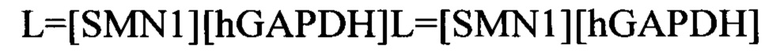
Экспрессию SMN1 после трансфекции рассчитывают с учетом фоновой экспрессии в контрольных клетках без добавления трансфецирующего агента и плазмид (КО):
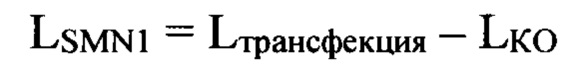
Критерии пригодности системы: эффективность прохождения ПЦР для каждой мишени находится в интервале 90-110%; значение R2 не ниже 0,99; количество детектируемых молекул превышает 10000/мкл реакционной смеси.
Определение белка SMN
Вестерн-блоттинг
10 мкл клеточного лизата инкубируют с 2,5 мкл загрузочного буфера с DTT LB, перемешивают и кипятят при 95°С 3 минуты. Проводят белковый электрофорез в полиакриламидном 12% геле. Режим проведения электрофореза для Glycine-SDS-PAGE: концентрирующий гель 70 В (20 мин, разделяющий гель 100-130 В, мин). Условия переноса: 1 час при 100 В. Для детекции белка SMN используют первичные антитела Anti-SMN/Gemin 1 antibody (ab223068) в разведении 1/5000 и вторичные антитела Goat Anti-Rabbit IgG H&L (HRP) в разведении 1/20 000. Для детекции бета-актина (контроль загрузки) используют первичные антитела β-Actin (8H10D10) Mouse mAb в разведении 1/1000 и вторичные антитела Goat Anti-Mouse IgG H&L (HRP) в разведении 1/20 000. Сигнал хемилюминесценции детектируют в гельдокументирующей системе Fusion SL2. Иммуноферментный анализ (ИФА)
Анализ проводят с помощью набора SMN ELISA Kit (Abcam©) по инструкции к набору. Перед анализом все реагенты, стрипы, стандартные и исследуемые образцы предварительно доводят до комнатной температуры. Перед каждым анализом проводят пробоподготовку стандартного образца SMN и образцов клеток. Стоковый раствор стандартного образца SMN с концентрацией 3 200 пг/мл готовят разведением 1 пробирки Standard в 1 мл Assay Buffer 13. Серией двукратных разведений в Assay Buffer 13 готовят растворы стандартов 1 600, 800, 400, 200, 100 и 50 пг/мл. В качестве бланкового образца с концентрацией SMN 0 пг/мл используют Assay Buffer 13. Клеточные лизаты разводят в Assay Buffer 13 до условной концентрации 500 и 2000 клеток/мл. Дополнительно оценивают однородность выделения белка с помощью набора DC protein assay (Bio-Rad) по инструкции к набору. Детектируют абсорбцию при основной длине волны 450 нм и референсной длине волны 580 нм на планшетном спектрофотометре. Проводят построение 4-х параметрической калибровочной кривой, используя значения, полученные для стандартных растворов. На основе калибровочной кривой прибор автоматически рассчитывает концентрации SMN в исследуемых образцах. Проводят сравнение пересчитанных концентраций SMN в пг/мл и абсолютных значений абсорбции в единицах оптической плотности.
Сборка и очистка рекомбинантных вирусных частиц AAV
Рекомбинантные вирусные частицы AAV получают посредством транзиентной трансфекции клеток НЕК293 тремя плазмидами:
1. Плазмидой ss-AAV-SMN1 или sc-AAV-SMN1 для экспрессии в составе одноцепочечного или самокомплементарного вектора соответственно, при этом кодируемый белок SMN может содержать аминокислотную последовательность, имеющую по меньшей мере 90% идентичность последовательности SEQ ID NO: 4;
2. Плазмидой для экспрессии генов репликации Rep серотипа AAV2, генов Сар серотипа AAV.PhP.B или AAV9, соответствующих SEQ ID NO: 1 и SEQ ID NO: 2 или с одиночными или несколькими точечными мутациями;
3. Плазмидой для экспрессии генов аденовируса Ad5, необходимых для сборки и упаковки капсидов AAV.
После суспезионного культивирования пакующих клеток НЕК293 и проведения трансфекции описанными плазмидами клетки лизируют, супернатант осветляют и концентрируют. После нескольких стадий хроматографической очистки целевой продукт обогащают методом ультрацентрифугирования в градиенте плотности иодиксанола (OptiPrep) или методом ионообменной хроматографии.
Титр вирусных частиц определяют с помощью методов количественной ПЦР.
Для приготовления препарата вирусных частиц используют раствор для формуляции, содержащий воду или буфер, хлористый натрий, глицин, гиалуроновую кислоту и/или другие эксципиенты. Финальный раствор вирусных частиц хранят замороженным (-20°С) или в лиофилизированной форме.
Пример 1. Получение генетической конструкции ss-AAV-SMN1
ПЦР-продукт размером 1 кб, полученный в результате амплификации плазмиды ATUM_126497_pF1257, содержащий последовательность гена SMN1 дикого типа SEQ ID NO: 1, с праймерами рг2324 (5'-ttccatcatcgat(ClaI)tgtaattcatgagccaccatggcgatgagcag-3') и pr2325 (5' -tagcggatctaga(XbaI)ttaatttaaggaatgtgagcaccttcc- 3'), вырезают из геля, очищают с использованием набора Cleanup Standard (Евроген). После инкубации с эндонуклеазами рестрикции ClaI и XbaI в течение 3-х часов при 37°С ДНК-фрагмент 1 очищают с помощью набора CleanMag DNA (Евроген). Вектор pAAV-GFP (Cell Biolabs) инкубируют с эндонуклеазами рестрикции ClaI и XbaI. Фрагмент размером 4,6 кб вырезают из геля, очищают с использованием набора Cleanup Standard (Евроген) и лигируют с ДНК-фрагментом 1 с помощью Т4 ДНК лигазы. Лигированную смесь используют для трансформации штамма с2523 (NEB) методом электропорации. Корректность плазмидной ДНК, выделенной из колоний, выросших на среде LB с ампицилином (100 мкг/мл), подтверждают секвенированием.
Финальная генетическая конструкция ss-AAV-SMN1 характеризуется следующими элементами, необходимыми для наработки плазмиды в Е. coli и сборке AAV в НЕК293:
1. Инвертированный тандемный повтор ITR, SEQ ID NO: 9;
2. CMV энхансер, SEQ ID NO: 10;
3. CMV промотер, SEQ ID NO: 11;
4. Интрон гена бета субъединицы гемоглобина, SEQ ID NO: 12;
5. SMN1, SEQ ID NO: 3;
6. Сигнал полиаденилирования hGH1, SEQ ID NO: 13;
7. Инвертированный тандемный повтор ITR, SEQ ID NO: 14;
8. Высоко-копийный бактериальный ориджин репликации
9. Ген устойчивости к ампицилину
Пример 2. Получение генетической конструкции sc-AAV-SMN1
На первом этапе получают конструкцию pF1254 путем замены гена устойчивости в плазмиде pscAAV-GFP addgene-323964 с ампицилиновой на канамициновую.
На следующем этапе получают конструкцию pF 1256 на основе плазмид плазмиды pF1254, в которую по сайтам рестрикции SacI и AvrII вставляют фрагмент ДНК, синтезированный компанией ATUM, в котором представлены промотор гена бета-актина курицы, интрон позднего гена 16S субъединицы вируса SV40 для конституитивной экспрессии; сигнал полиаденилирования BGH.
Далее получают конструкцию pF1262 на основе плазмид addgene-plasmid-92399-sequence-178249 и pF1256. ПЦР-продукт размером 859 п. о., полученный в результате амплификации плазмиды addgene-plasmid-92399-sequence-178249 с праймерами Pr2344 (5' - cccaactgatcttcagcatc - 3') и Pr2337 (5'- cgcgcttcgctttttatagg - 3'), вырезают из геля, очищают с использованием набора Cleanup Standard (Евроген). После инкубации с эндонуклеазами рестрикции SpeI и EciI в течение 3-х часов при 37°С ДНК-фрагмент 1 очищают с помощью набора CleanMag DNA (Евроген). Вектор pF1256 инкубируют с эндонуклеазами рестрикции SpeI и BsrDI. Фрагмент размером 2,9 кб вырезают из геля, очищают с использованием набора Cleanup Standard (Евроген) и лигируют с ДНК-фрагментом 1 с помощью Т4 ДНК лигазы. Лигированную смесь используют для трансформации штамма Е. coli методом электропорации. Корректность плазмидной ДНК, выделенной из колоний, выросших на среде LB с канамицином (50 мкг/мл), определяют секвенированием.
Для создания генетической конструкции sc-AAV-SMN1 вектор, полученный на предыдущем этапе, и ATUM_126497_pF1257 инкубируют с эндонуклеазами рестрикции EcoRI-HF и XbaI. Соответствующие фрагменты размерами 3,3 кб и 1 кб вырезают из геля, очищают с использованием набора Cleanup Standard (Евроген) и лигируют с помощью Т4 ДНК лигазы. Лигированную смесь используют для трансформации штамма Е. coli методом электропорации. Корректность плазмидной ДНК, выделенной из колоний, выросших на среде LB с канамицином (50 мкг/мл), определяют секвенированием.
Финальная генетическая конструкция sc-AAV-SMN1 характеризуется следующими элементами, необходимыми для наработки плазмиды в Е. coli и сборке AAV в НЕК293:
1. модифицированный инвертированный тандемный повтор ITR-dTRS, SEQ ID NO: 15;
2. CMV энхансер, SEQ ID NO: 16;
3. промотор гена бета-актина курицы, SEQ ID NO: 17;
4. интрон позднего гена 16S субъединицы вируса SV40, SEQ ID NO: 18;
5. SMN1, SEQ ID NO: 3;
6. сигнал полиаденилирования BGH, SEQ ID NO: 19;
7. инвертированный тандемный повтор ITR, SEQ ID NO: 20;
8. высоко-копийный бактериальный ориджин репликации
9. ген устойчивости к канамицину
Пример 3. Проверка экспрессии гена SMN1 после трансфекции плазмидами sc-AAV-SMN1 и ss-AAV-SMN1
На фиг. 1А и фиг. 1 В представлены результаты анализа экспрессии SMN1 в образцах методом ОТ-ПЦР в реальном времени. В исследуемых образцах после трансфекции наблюдается экспрессия SMN1, в то время как для образцов отрицательного контроля без добавления плазмид сигнал остается на фоновом уровне. При этом во всех исследуемых образцах наблюдается экспрессия гена домашнего хозяйства hGAPDH на сопоставимом уровне, что потдверждает качество пробоподготовки и однородность загрузки образцов для проведения ПЦР реакции.
Во всех образцах трансфецированных плазмидами наблюдается экспрессия гена SMN1. При этом уровень мРНК SMN1 в 4 раза превышает уровень экспрессии для экспрессионной конструкции ss-AAV-SMN1 по сравнению с sc-AAV-SMN1.
Пример 4. Проверка трансляции белка SMN после трансфекции плазмидами sc-AAV-SMN1 и ss-AAV-SMN1
На фиг. 2А представлены результаты определения белка SMN в клеточных лизатах после трансфекции методом Вестерн-блоттинга. После инкубации с антителами, узнающими белок SMN, наблюдается специфичная полоса размера, соответствующего по молекулярной массе целевому белку (31кДа SMN), в контрольных образцах без трансфекции сигнал отсутствует. Инкубация с антителами, узнающими ген домашнего хозяйства актин, подтверждает однородность загрузки образцов. Полученные данные подтверждают наличие белка SMN в исследуемых клеточных лизатах.
Содержание белка SMN в трансфецированных клетках было количественно определено с помощью иммуноферментного анализа (фиг. 2 В).
Для образцов после трансфекции плазмидами ss-AAV-SMN1 и sc-AAV-SMN1 наблюдается достоверное отличие от КО, при этом сигнал для ss-AAV-SMN1 превышает сигнал для sc-AAV-SMN1 в 6,3 раза.
Таким образом, экспрессия гена SMN1 на уровне мРНК (фиг. 1) соответствуют уровням трансляции белка SMN (фиг. 2).
Пример 5. Получение генетической конструкции для экспрессии белков Rep (AAV2) и Cap (AAV-PhP.B)
Для получения генетической конструкции Rep2 PhP.B коммерческие вектора pUCmini-iCAP-PHP.B (Addgene, 103002) и pAAV2-9n (Addgene, 112865) инкубируют с эндонуклеазами рестрикции Sbf1 и BsiWI. Фрагменты размерами 1,2 кб (pUCmini-iCAP-РНР.В) и 6 кб (pAAV2-9n) вырезают из геля, очищают с использованием набора Cleanup Standard (Евроген) и лигируют с помощью Т4 ДНК лигазы. Лигированную смесь используют для трансформации штамма с2523 (NEB) методом электропорации. Корректность плазмидной ДНК, выделенной из колоний, выросших на среде LB с ампицилином (100 мкг/мл), подтверждают секвенированием.
Пример 6. Создание вирусных препаратов ss-AAV и sc-AAV, несущих функциональный ген SMN1
Для трансфекции пакующих клеток НЕК293 использовались следующие генетические конструкции:
1. Плазмида ss-AAV-SMN1 или sc-AAV-SMN1 для экспрессии гена SMN1 в составе одноцепочечного или самокомплементарного вектора согласно SEQ ID NO: 5 и SEQ ID NO: 6 соответственно;
2. Плазмида для экспрессии генов репликации Rep серотипа AAV2, генов Сар серотипа AAVPhP.B или AAV9, соответствующих SEQ ID NO: 7 и SEQ ID NO: 8;
3. Плазмида для экспрессии генов аденовируса Ad5.
Суспезионное культивирование пакующих клеток НЕК293 проводили в волновом биореакторе, через 72 часа после трансфекции клетки лизировали добавлением дететергента Triton X100, затем осуществляли глубинную фильтрацию полученного лизата через фильтр Clarisolve 20MS или аналогичный. Затем проводили фильтрацию с использованием мембранного капсульного гидрофильного фильтра с размером пор 0,2 um, концетрирование и диафильтрацию с помощью TFF. На первой стадии хроматографической очистки использовали сорбент POROS CaptureSelect AAV9 Affinity Resin. Далее проводили ионно-обменную хроматографию с использованием колонки CIMmultus® QA 1 mL Monolithic Column (2 μm) или аналогичной, предназначенной для разделения больших биомолекул. На последнем этапе осуществляли замену буфера с помощью TFF. В качестве буфера для формуляци использовали PBS, рН 7,4 с добавлением 0,005% Pluronic F68 для снижения агрегации вирусных частиц.
Пример 7. Эффект конструкций на выживаемость мышей SMNA7
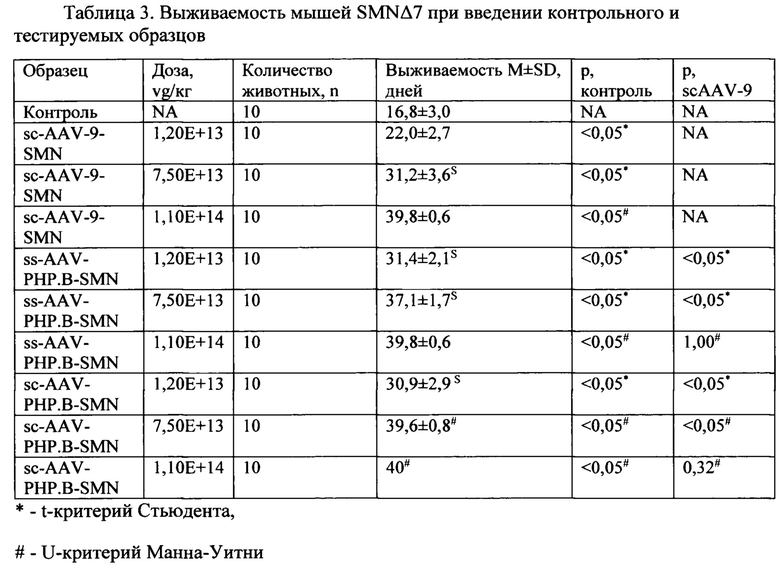
Настоящее исследование проведено с использованием линий мышей FVB/N SMNΔ7. Данная линия лишена эндогенного мышиного гена SMN1, но имеет две копии интактного человеческого SMN2 (hSMN2) и еще две копии hSMN2 с удаленным экзоном 7, которые вместе обеспечивают достаточную экспрессию SMN для предотвращения эмбриональной гибели. Модель на основе линии мышей FVB/N SMNA7 воспроизводит такие особенности течения спинальной мышечной атрофии (СМА), как низкий уровень экспрессии SMN, потеря двигательных нейронов, слабость и преждевременная смерть. Поставщик животных - JAX®, strain #005025. По данным исследований лаборатории выживаемость гомозигот составляет 15-22 дня. Выживаемость животных в течение 40 дней и более соответствует полному замещению функции гена SMN1 у мышей FVB/N SMNA7.
Исследуемые образцы вводили новорожденным мышам внутривенно в височную вену. Для каждого образца было исследовано по три дозы: 1,20Е+13, 7,50Е+13 vg/кг и 1,10Е+14 vg/кг, в качестве контроля вводили фосфатно-солевой буфер с добавлением 0,005% Pluronic F68. В качестве тестируемых образцов были исследованы конструкции на основе серотипа sc-AAV-9, ss-AAV-PHP.B и sc-AAV-PHP.B. Все конструкции были получены способом, указанным выше. После введения тестируемых или контрольного образцов за животными наблюдали в течение 40 дней.
Для значения выживаемости приведена описательная статистика в виде среднего (Mean) и стандартного отклонения (SD) в соответствии с рекомендациями Руководства по проведению доклинических исследований лекарственных средств [18]. Оценка нормальности распределения выполнена с помощью теста Шапиро-Уилка. Для межгрупповых сравнений использовали t-критерий Стьюдента или U-критерия Манна-Уитни в зависимости от результатов теста на нормальность распределения. Различия считали достоверными при р<0,05. Все тестируемые образцы сравнивали с контролем, образцы на основе капсида AAV-PHP.B также сравнивали с аналогичными дозами образцов на основе капсида AAV-9.
По результатам исследования выживаемость животных в группах, которые получили тестируемые образцы, была значимо выше, чем в контрольной группе (Таблица 3). Попарное сравнение аналогичных доз конструкций на основе AAV-PHP.B и AAV-9 продемонстрировало значимое превосходство капсида AAV-PHP.B при введении препарата в дозах ниже 1,10Е+14 vg/кг. Наибольшая эффективность была зарегистрирована для конструкции на основе вирусного вектора sc-AAV-PHP.B-SMN: данная композиция в дозе 7,50Е+13 vg/кг по эффективности была сопоставима с дозой 1,10Е+14 vg/кг для sc-AAV-9-SMN. Использование AAV-PHP.B в качестве вектора для доставки гена SMN1 способствует достижению целевой выживаемости при введении меньших доз препарата вероятно вследствие более эффективной трансдукции целевой популяции клеток.
Таким образом, благодаря увеличенной проницаемости гематоэнцефалического барьера и селективности трансдукции для конструкций на основе вектора AAV-PHP.B возможен более благоприятный профиль безопасности препарата для лечения спинальной мышечной атрофии. Применение конструкций на основе ss-AAV-PHP.B может способствовать лучшему профилю безопасности при использовании аналогичных доз с sc-AAV-9 из-за потенциально сниженной иммуногенности.
Использование серотипа AAV-PHP.B может способствовать уменьшению дозы по сравнению с уже зарегистрированными препаратами генной терапии и/или уменьшить частоту нежелательных реакций на введение препарата. Кроме того, возможное снижение дозы и/или улучшение профиля безопасности позволят применять данный препарат у пациентов старше двух лет.
Список литературы
1. Markowitz J.A., Singh P., Darras В.Т. Spinal Muscular Atrophy: A Clinical and Research Update. Pediatric Neurology. 2012, 46(1): 1-12. doi:10.1016/j.pediatrneurol.2011.09.001
2. Mercuri E., Finkel R. S., Muntoni F. et al. Diagnosis and management of spinal muscular atrophy: Part 1: Recommendations for diagnosis, rehabilitation, orthopedic and nutritional care. Neuromuscular Disorders. 2018, 2:103-115. https://doi.Org/10.1016/j.nmd.2017.11.005
3. Butchbach M. E. Copy Number Variations in the Survival Motor Neuron Genes: Implications for Spinal Muscular Atrophy and Other Neurodegenerative Diseases. Frontiers in Molecular Biosciences. 2016; 3:7. doi:10.3389/fmolb.2016.00007
4. Prior T.W., Leach M.E., Finanger E. Spinal Muscular Atrophy. 2000 Feb 24 [Updated 2020 Dec 3]. In: Adam M.P., Ardinger H.H, Pagon R.A, et al., editors. GeneReviews® [Internet]. Seattle (WA): University of Washington, Seattle; 1993-2021. Available from: https://www.ncbi.nlm.nih.gov/books/NBK1352/
5. Passini M.A., Cheng S.H. Prospects for the gene therapy of spinal muscular atrophy. Trends in Molecular Medicine, 2011; 17(5):259-265. doi: 10.1016/j.molmed.2011.01.002
6. Food and Drug Administration (FDA). URL: https://www.fda.gov/vaccines-blood-biologics/zolgensma (дата обращения 04.05.22)
7. Joseph R. Smith-Arica, Jeffrey S. Bartlett. Gene therapy: Recombinant adeno-associated virus vectors. Current Cardiology Reports. 2001; 3:43-49.
8. Passini M.A., Lee E.В., Heuer H.H., Wolf J.H. Distribution of a Lysosomal Enzyme in the Adult Brain by Axonal Transport and by Cells of the Rostral Migratory Stream. The Journal of Neuroscience, 2002; 22:6437-6446. doi:10.1523/JNEUROSCI.22-15-06437.2002
9. High K. A., Aubourg P. rAAV human trial experience. Methods in Molecular Biology. 2011; 807:429-457.
10. Xie Q., Bu W., Bhatia S., Hare J., Somasundaram Т., Azzi A., Chapman M.S. The atomic structure of adeno-associated virus (AAV-2), a vector for human gene therapy. Proceedings of the National Academy of Sciences, 2002; 99(16), 10405-10410. doi: 10.1073/pnas. 162250899
11. McCarty D.M. Self-complementary AAV Vectors; Advances and Applications. Molecular Therapy. 2008; 16(10): 1648-1656. doi:10.1038/mt.2008.171
12. Martino A.T, Suzuki M, Markusic D.M, et al. The genome of self-complementary adeno-associated viral vectors increases Toll-like receptor 9-dependent innate immune responses in the liver. Blood. 2011;117(24):6459-6468. doi:10.1182/blood-2010-10-314518
13. Food and Drug Administration (FDA). URL: https://www.fda.gov/media/126109/download (дата обращения 04.05.22)
14. Food and Drug Administration (FDA). Cellular, Tissue, and Gene Therapies Advisory Committee (CTGTAC) Meeting #70. Toxicity Risks of Adeno-associated Virus (AAV) Vectors for Gene Therapy (GT), September 2-3, 2021. URL: https://www.fda.gov/media/151599/download_(дата обращения 04.05.22).
15. BLA Clinical Review Memorandum, onasemnogene abeparvovec-xioi, STN 125694/0, May 23, 2019. URL: https://www.fda.gov/media/128116/download (дата обращения 04.05.22).
16. Deverman В.E., Pravdo P.L., Simpson B.P., Kumar S.R., Chan K.Y., Banerjee A., Gradinaru V. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 2016, 34(2): 204-209. doi:10.1038/nbt.3440
17. Sambrook J. F. Molecular Cloning: a Laboratory Manual (Fourth Edition). Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press, 2014.
18. Руководство по проведению доклинических исследований лекарственных средств. Часть первая. М.: Гриф и К, 2012.
--->
<ST26SequenceListing dtdVersion="V1_3"
fileName="SMA_gene_therapy_2022_05_06.xml" softwareName="WIPO
Sequence" softwareVersion="1.1.0" productionDate="2022-05-06">
<ApplicantFileReference>5-D</ApplicantFileReference>
<ApplicantName languageCode="ru">ГЕРОФАРМ</ApplicantName>
<ApplicantNameLatin>GEROPHARM</ApplicantNameLatin>
<InventorName languageCode="ru">Родионов Петр Петрович</InventorName>
<InventorNameLatin>Rodionov Petr Petrovich</InventorNameLatin>
<InventionTitle languageCode="ru">Аденоассоциированный вирусный
вектор, состоящий из белков капсида PHP.B, нуклеиновой кислоты,
кодирующей белок SMN, и его применение</InventionTitle>
<SequenceTotalQuantity>22</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>743</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SOURCE</INSDFeature_key>
<INSDFeature_location>1..743</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>MOL_TYPE</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>ORGANISM</INSDQualifier_name>
<INSDQualifier_value>Adeno-associated dependoparvovirus
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MAADGYLPDWLEDNLSEGIREWWALKPGAPQPKANQQHQDNARGLVLPGYKY
LGPGNGLDKGEPVNAADAAALEHDKAYDQQLKAGDNPYLKYNHADAEFQERLKEDTSFGGNLGRAVFQAK
KRLLEPLGLVEEAAKTAPGKKRPVEQSPQEPDSSAGIGKSGAQPAKKRLNFGQTGDTESVPDPQPIGEPP
AAPSGVGSLTMASGGGAPVADNNEGADGVGSSSGNWHCDSQWLGDRVITTSTRTWALPTYNNHLYKQISN
STSGGSSNDNAYFGYSTPWGYFDFNRFHCHFSPRDWQRLINNNWGFRPKRLNFKLFNIQVKEVTDNNGVK
TIANNLTSTVQVFTDSDYQLPYVLGSAHEGCLPPFPADVFMIPQYGYLTLNDGSQAVGRSSFYCLEYFPS
QMLRTGNNFQFSYEFENVPFHSSYAHSQSLDRLMNPLIDQYLYYLSRTINGSGQNQQTLKFSVAGPSNMA
VQGRNYIPGPSYRQQRVSTTVTQNNNSEFAWPGASSWALNGRNSLMNPGPAMASHKEGEDRFFPLSGSLI
FGKQGTGRDNVDADKVMITNEEEIKTTNPVATESYGQVATNHQSAQTLAVPFKAQAQTGWVQNQGILPGM
VWQDRDVYLQGPIWAKIPHTDGNFHPSPLMGGFGMKHPPPQILIKNTPVPADPPTAFNKDKLNSFITQYS
TGQVSVEIEWELQKENSKRWNPEIQYTSNYYKSNNVEFAVNTEGVYSEPRPIGTRYLTRNL</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>736</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SOURCE</INSDFeature_key>
<INSDFeature_location>1..736</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>MOL_TYPE</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>ORGANISM</INSDQualifier_name>
<INSDQualifier_value>Adeno-associated dependoparvovirus
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MAADGYLPDWLEDNLSEGIREWWALKPGAPQPKANQQHQDNARGLVLPGYKY
LGPGNGLDKGEPVNAADAAALEHDKAYDQQLKAGDNPYLKYNHADAEFQERLKEDTSFGGNLGRAVFQAK
KRLLEPLGLVEEAAKTAPGKKRPVEQSPQEPDSSAGIGKSGAQPAKKRLNFGQTGDTESVPDPQPIGEPP
AAPSGVGSLTMASGGGAPVADNNEGADGVGSSSGNWHCDSQWLGDRVITTSTRTWALPTYNNHLYKQISN
STSGGSSNDNAYFGYSTPWGYFDFNRFHCHFSPRDWQRLINNNWGFRPKRLNFKLFNIQVKEVTDNNGVK
TIANNLTSTVQVFTDSDYQLPYVLGSAHEGCLPPFPADVFMIPQYGYLTLNDGSQAVGRSSFYCLEYFPS
QMLRTGNNFQFSYEFENVPFHSSYAHSQSLDRLMNPLIDQYLYYLSKTINGSGQNQQTLKFSVAGPSNMA
VQGRNYIPGPSYRQQRVSTTVTQNNNSEFAWPGASSWALNGRNSLMNPGPAMASHKEGEDRFFPLSGSLI
FGKQGTGRDNVDADKVMITNEEEIKTTNPVATESYGQVATNHQSAQAQAQTGWVQNQGILPGMVWQDRDV
YLQGPIWAKIPHTDGNFHPSPLMGGFGMKHPPPQILIKNTPVPADPPTAFNKDKLNSFITQYSTGQVSVE
IEWELQKENSKRWNPEIQYTSNYYKSNNVEFAVNTEGVYSEPRPIGTRYLTRNL</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>885</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..885</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggcgatgagcagcggcggcagtggtggcggcgtcccggagcaggaggatt
ccgtgctgttccggcgcggcacaggccagagcgatgattctgacatttgggatgatacagcactgataaa
agcatatgataaagctgtggcttcatttaagcatgctctaaagaatggtgacatttgtgaaacttcgggt
aaaccaaaaaccacacctaaaagaaaacctgctaagaagaataaaagccaaaagaagaatactgcagctt
ccttacaacagtggaaagttggggacaaatgttctgccatttggtcagaagacggttgcatttacccagc
taccattgcttcaattgattttaagagagaaacctgtgttgtggtttacactggatatggaaatagagag
gagcaaaatctgtccgatctactttccccaatctgtgaagtagctaataatatagaacagaatgctcaag
agaatgaaaatgaaagccaagtttcaacagatgaaagtgagaactccaggtctcctggaaataaatcaga
taacatcaagcccaaatctgctccatggaactcttttctccctccaccaccccccatgccagggccaaga
ctgggaccaggaaagccaggtctaaaattcaatggcccaccaccgccaccgccaccaccaccaccccact
tactatcatgctggctgcctccatttccttctggaccaccaataattcccccaccacctcccatatgtcc
agattctcttgatgatgctgatgctttgggaagtatgttaatttcatggtacatgagtggctatcatact
ggctattatatgggttttagacaaaatcaaaaagaaggaaggtgctcacattccttaaattaa</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>294</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SOURCE</INSDFeature_key>
<INSDFeature_location>1..294</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>MOL_TYPE</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>ORGANISM</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MAMSSGGSGGGVPEQEDSVLFRRGTGQSDDSDIWDDTALIKAYDKAVASFKH
ALKNGDICETSGKPKTTPKRKPAKKNKSQKKNTAASLQQWKVGDKCSAIWSEDGCIYPATIASIDFKRET
CVVVYTGYGNREEQNLSDLLSPICEVANNIEQNAQENENESQVSTDESENSRSPGNKSDNIKPKSAPWNS
FLPPPPPMPGPRLGPGKPGLKFNGPPPPPPPPPPHLLSCWLPPFPSGPPIIPPPPPICPDSLDDADALGS
MLISWYMSGYHTGYYMGFRQNQKEGRCSHSLN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>2927</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2927</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q41">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctgcaggcagctgcgcgctcgctcgctcactgaggccgcccgggcgtcggg
cgacctttggtcgcccggcctcagtgagcgagcgagcgcgcagagagggagtggccaactccatcactag
gggttcctgcggccgcacgcgtggagctagttattaatagtaatcaattacggggtcattagttcatagc
ccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaacgaccccc
gcccattgacgtcaataatgacgtatgttcccatagtaacgtcaatagggactttccattgacgtcaatg
ggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgccaagtacgccccct
attgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttccta
cttggcagtacatctacgtattagtcatcgctattaccatggtgatgcggttttggcagtacatcaatgg
gcgtggatagcggtttgactcacggggatttccaagtctccaccccattgacgtcaatgggagtttgttt
tgcaccaaaatcaacgggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtag
gcgtgtacggtgggaggtctatataagcagagctcgtttagtgaaccgtcagatcgcctggagacgccat
ccacgctgttttgacctccatagaagacaccgggaccgatccagcctccgcggattcgaatcccggccgg
gaacggtgcattggaacgcggattccccgtgccaagagtgacgtaagtaccgcctatagagtctataggc
ccacaaaaaatgctttcttcttttaatatacttttttgtttatcttatttctaatactttccctaatctc
tttctttcagggcaataatgatacaatgtatcatgcctctttgcaccattctaaagaataacagtgataa
tttctgggttaaggcaatagcaatatttctgcatataaatatttctgcatataaattgtaactgatgtaa
gaggtttcatattgctaatagcagctacaatccagctaccattctgcttttattttatggttgggataag
gctggattattctgagtccaagctaggcccttttgctaatcatgttcatacctcttatcttcctcccaca
gctcctgggcaacgtgctggtctgtgtgctggcccatcactttggcaaagaattgggattcgaacatcga
ttgtaattcatgagccaccatggcgatgagcagcggcggcagtggtggcggcgtcccggagcaggaggat
tccgtgctgttccggcgcggcacaggccagagcgatgattctgacatttgggatgatacagcactgataa
aagcatatgataaagctgtggcttcatttaagcatgctctaaagaatggtgacatttgtgaaacttcggg
taaaccaaaaaccacacctaaaagaaaacctgctaagaagaataaaagccaaaagaagaatactgcagct
tccttacaacagtggaaagttggggacaaatgttctgccatttggtcagaagacggttgcatttacccag
ctaccattgcttcaattgattttaagagagaaacctgtgttgtggtttacactggatatggaaatagaga
ggagcaaaatctgtccgatctactttccccaatctgtgaagtagctaataatatagaacagaatgctcaa
gagaatgaaaatgaaagccaagtttcaacagatgaaagtgagaactccaggtctcctggaaataaatcag
ataacatcaagcccaaatctgctccatggaactcttttctccctccaccaccccccatgccagggccaag
actgggaccaggaaagccaggtctaaaattcaatggcccaccaccgccaccgccaccaccaccaccccac
ttactatcatgctggctgcctccatttccttctggaccaccaataattcccccaccacctcccatatgtc
cagattctcttgatgatgctgatgctttgggaagtatgttaatttcatggtacatgagtggctatcatac
tggctattatatgggttttagacaaaatcaaaaagaaggaaggtgctcacattccttaaattaatctaga
gtcgacctgcagaagcttgcctcgagcagcgctgctcgagagatctacgggtggcatccctgtgacccct
ccccagtgcctctcctggccctggaagttgccactccagtgcccaccagccttgtcctaataaaattaag
ttgcatcattttgtctgactaggtgtccttctataatattatggggtggaggggggtggtatggagcaag
gggcaagttgggaagacaacctgtagggcctgcggggtctattgggaaccaagctggagtgcagtggcac
aatcttggctcactgcaatctccgcctcctgggttcaagcgattctcctgcctcagcctcccgagttgtt
gggattccaggcatgcatgaccaggctcagctaatttttgtttttttggtagagacggggtttcaccata
ttggccaggctggtctccaactcctaatctcaggtgatctacccaccttggcctcccaaattgctgggat
tacaggcgtgaaccactgctcccttccctgtccttctgattttgtaggtaaccacgtgcggaccgagcgg
ccgcaggaacccctagtgatggagttggccactccctctctgcgcgctcgctcgctcactgaggccgggc
gaccaaaggtcgcccgacgcccgggctttgcccgggcggcctcagtgagcgagcgagcgcgcagctgcct
gcagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>2405</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2405</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccctctctgcgcgctcgctcgctcactgaggccgcccgggcaaagcccggg
cgtcgggcgacctttggtcgcccggcctcagtgagcgagcgagcgcgcagagagggagtggactagttat
taatagtaatcaattacggggtcattagttcatagcccatatatggagttccgcgttacataacttacgg
taaatggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgtatgttcccat
agtaacgccaatagggactttccattgacgtcaatgggtggagtatttacggtaaactgcccacttggca
gtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctggc
attatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctat
taccatggtcgaggtgagccccacgttctgcttcactctccccatctcccccccctccccacccccaatt
ttgtatttatttattttttaattattttgtgcagcgatgggggcggggggggggggggcgcgcgccaggc
ggggcggggcggggcgaggggcggggcggggcgaggcggagaggtgcggcggcagccaatcagagcggcg
cgctccgaaagtttccttttatggcgaggcggcggcggcggcggccctataaaaagcgaagcgcgcggcg
ggcgggagcgggatcagccaccgcggtggcggcctagagtcgacgaggaactgaaaaaccagaaagttaa
ctggtaagtttagtctttttgtcttttatttcaggtcccggatccggtggtggtgcaaatcaaagaactg
ctcctcagtggatgttgcctttacttctaggcctgtacggaagtgttacttctgctctaaaagctgcgga
attgtacccgcggccgatccaccggtccggaattcccgggatatcccacgctagcggccccacgctgcgc
acccgcgggtttgctatggcgatgagcagcggcggcagtggtggcggcgtcccggagcaggaggattccg
tgctgttccggcgcggcacaggccagagcgatgattctgacatttgggatgatacagcactgataaaagc
atatgataaagctgtggcttcatttaagcatgctctaaagaatggtgacatttgtgaaacttcgggtaaa
ccaaaaaccacacctaaaagaaaacctgctaagaagaataaaagccaaaagaagaatactgcagcttcct
tacaacagtggaaagttggggacaaatgttctgccatttggtcagaagacggttgcatttacccagctac
cattgcttcaattgattttaagagagaaacctgtgttgtggtttacactggatatggaaatagagaggag
caaaatctgtccgatctactttccccaatctgtgaagtagctaataatatagaacagaatgctcaagaga
atgaaaatgaaagccaagtttcaacagatgaaagtgagaactccaggtctcctggaaataaatcagataa
catcaagcccaaatctgctccatggaactcttttctccctccaccaccccccatgccagggccaagactg
ggaccaggaaagccaggtctaaaattcaatggcccaccaccgccaccgccaccaccaccaccccacttac
tatcatgctggctgcctccatttccttctggaccaccaataattcccccaccacctcccatatgtccaga
ttctcttgatgatgctgatgctttgggaagtatgttaatttcatggtacatgagtggctatcatactggc
tattatatgggttttagacaaaatcaaaaagaaggaaggtgctcacattccttaaattaaggagaaatgc
tggcatagagcagcactaaatgacaccactaaagaaacgatcagacagatctagataggtaccgctgatc
agcctcgactgtgccttctagttgccagccatctgttgtttgcccctcccccgtgccttccttgaccctg
gaaggtgccactcccactgtcctttcctaataaaatgaggaaattgcatcgcattgtctgagtaggtgtc
attctattctggggggtggggtggggcaggacagcaagggggaggattgggaagacaatagcaggcatgc
tggggagagatcgatctgcctaggtgcggaccgagcggccgcaggaacccctagtgatggagttggccac
tccctctctgcgcgctcgctcgctcactgaggccgggcgaccaaaggtcgcccgacgcccgggctttgcc
cgggcggcctcagtgagcgagcgagcgcgcagctgcctgcagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>2232</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2232</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Adeno-associated dependoparvovirus
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggctgccgatggttatcttccagattggctcgaggacaaccttagtgaag
gaattcgcgagtggtgggctttgaaacctggagcccctcaacccaaggcaaatcaacaacatcaagacaa
cgctcgaggtcttgtgcttccgggttacaaataccttggacccggcaacggactcgacaagggggagccg
gtcaacgcagcagacgcggcggccctcgagcacgacaaggcctacgaccagcagctcaaggccggagaca
acccgtacctcaagtacaaccacgccgacgccgagttccaggagcggctcaaagaagatacgtcttttgg
gggcaacctcgggcgagcagtcttccaggccaaaaagaggcttcttgaacctcttggtctggttgaggaa
gcggctaagacggctcctggaaagaagaggcctgtagagcagtctcctcaggaaccggactcctccgcgg
gtattggcaaatcgggtgcacagcccgctaaaaagagactcaatttcggtcagactggcgacacagagtc
agtcccagaccctcaaccaatcggagaacctcccgcagccccctcaggtgtgggatctcttacaatggct
tcaggtggtggcgcaccagtggcagacaataacgaaggtgccgatggagtgggtagttcctcgggaaatt
ggcattgcgattcccaatggctgggggacagagtcatcaccaccagcacccgaacctgggccctgcccac
ctacaacaatcacctctacaagcaaatctccaacagcacatctggaggatcttcaaatgacaacgcctac
ttcggctacagcaccccctgggggtattttgacttcaacagattccactgccacttctcaccacgtgact
ggcagcgactcatcaacaacaactggggattccggcctaagcgactcaacttcaagctcttcaacattca
ggtcaaagaggttacggacaacaatggagtcaagaccatcgccaataaccttaccagcacggtccaggtc
ttcacggactcagactatcagctcccgtacgtgctcgggtcggctcacgagggctgcctcccgccgttcc
cagcggacgttttcatgattcctcagtacgggtatctgacgcttaatgatggaagccaggccgtgggtcg
ttcgtccttttactgcctggaatatttcccgtcgcaaatgctaagaacgggtaacaacttccagttcagc
tacgagtttgagaacgtacctttccatagcagctacgctcacagccaaagcctggaccgactaatgaatc
cactcatcgaccaatacttgtactatctctctagaactattaacggttctggacagaatcaacaaacgct
aaaattcagtgtggccggacccagcaacatggctgtccagggaagaaactacatacctggacccagctac
cgacaacaacgtgtctcaaccactgtgactcaaaacaacaacagcgaatttgcttggcctggagcttctt
cttgggctctcaatggacgtaatagcttgatgaatcctggacctgctatggcctctcacaaagaaggaga
ggaccgtttctttcctttgtctggatctttaatttttggcaaacaaggtactggcagagacaacgtggat
gcggacaaagtcatgataaccaacgaagaagaaattaaaactactaacccggtagcaacggagtcctatg
gacaagtggccacaaaccaccagagtgcccaaactttggcggtgccttttaaggcacaggcgcagaccgg
ttgggttcaaaaccaaggaatacttccgggtatggtttggcaggacagagatgtgtacctgcaaggaccc
atttgggccaaaattcctcacacggacggcaactttcacccttctccgctgatgggagggtttggaatga
agcacccgcctcctcagatcctcatcaaaaacacacctgtacctgcggatcctccaacggccttcaacaa
ggacaagctgaactctttcatcacccagtattctactggtcaagtcagcgtggagatcgagtgggagctg
cagaaggaaaacagcaagcgctggaacccggagatccagtacacttccaactattacaagtctaataatg
ttgaatttgctgttaatactgaaggtgtatatagtgaaccccgccccattggcaccagatacctgactcg
taatctgtaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>2211</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2211</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Adeno-associated dependoparvovirus
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggctgccgatggttatcttccagattggctcgaggacaaccttagtgaag
gaattcgcgagtggtgggctttgaaacctggagcccctcaacccaaggcaaatcaacaacatcaagacaa
cgctcgaggtcttgtgcttccgggttacaaataccttggacccggcaacggactcgacaagggggagccg
gtcaacgcagcagacgcggcggccctcgagcacgacaaggcctacgaccagcagctcaaggccggagaca
acccgtacctcaagtacaaccacgccgacgccgagttccaggagcggctcaaagaagatacgtcttttgg
gggcaacctcgggcgagcagtcttccaggccaaaaagaggcttcttgaacctcttggtctggttgaggaa
gcggctaagacggctcctggaaagaagaggcctgtagagcagtctcctcaggaaccggactcctccgcgg
gtattggcaaatcgggtgcacagcccgctaaaaagagactcaatttcggtcagactggcgacacagagtc
agtcccagaccctcaaccaatcggagaacctcccgcagccccctcaggtgtgggatctcttacaatggct
tcaggtggtggcgcaccagtggcagacaataacgaaggtgccgatggagtgggtagttcctcgggaaatt
ggcattgcgattcccaatggctgggggacagagtcatcaccaccagcacccgaacctgggccctgcccac
ctacaacaatcacctctacaagcaaatctccaacagcacatctggaggatcttcaaatgacaacgcctac
ttcggctacagcaccccctgggggtattttgacttcaacagattccactgccacttctcaccacgtgact
ggcagcgactcatcaacaacaactggggattccggcctaagcgactcaacttcaagctcttcaacattca
ggtcaaagaggttacggacaacaatggagtcaagaccatcgccaataaccttaccagcacggtccaggtc
ttcacggactcagactatcagctcccgtacgtgctcgggtcggctcacgagggctgcctcccgccgttcc
cagcggacgttttcatgattcctcagtacgggtatctgacgcttaatgatggaagccaggccgtgggtcg
ttcgtccttttactgcctggaatatttcccgtcgcaaatgctaagaacgggtaacaacttccagttcagc
tacgagtttgagaacgtacctttccatagcagctacgctcacagccaaagcctggaccgactaatgaatc
cactcatcgaccaatacttgtactatctctcaaagactattaacggttctggacagaatcaacaaacgct
aaaattcagtgtggccggacccagcaacatggctgtccagggaagaaactacatacctggacccagctac
cgacaacaacgtgtctcaaccactgtgactcaaaacaacaacagcgaatttgcttggcctggagcttctt
cttgggctctcaatggacgtaatagcttgatgaatcctggacctgctatggccagccacaaagaaggaga
ggaccgtttctttcctttgtctggatctttaatttttggcaaacaaggaactggaagagacaacgtggat
gcggacaaagtcatgataaccaacgaagaagaaattaaaactactaacccggtagcaacggagtcctatg
gacaagtggccacaaaccaccagagtgcccaagcacaggcgcagaccggctgggttcaaaaccaaggaat
acttccgggtatggtttggcaggacagagatgtgtacctgcaaggacccatttgggccaaaattcctcac
acggacggcaactttcacccttctccgctgatgggagggtttggaatgaagcacccgcctcctcagatcc
tcatcaaaaacacacctgtacctgcggatcctccaacggccttcaacaaggacaagctgaactctttcat
cacccagtattctactggccaagtcagcgtggagatcgagtgggagctgcagaaggaaaacagcaagcgc
tggaacccggagatccagtacacttccaactattacaagtctaataatgttgaatttgctgttaatactg
aaggtgtatatagtgaaccccgccccattggcaccagatacctgactcgtaatctgtaa</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>130</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..130</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Adeno-associated dependoparvovirus
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctgcaggcagctgcgcgctcgctcgctcactgaggccgcccgggcgtcggg
cgacctttggtcgcccggcctcagtgagcgagcgagcgcgcagagagggagtggccaactccatcactag
gggttcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>304</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..304</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Human betaherpesvirus 5</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccc
cgcccattgacgtcaataatgacgtatgttcccatagtaacgtcaatagggactttccattgacgtcaat
gggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgccaagtacgccccc
tattgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttcct
acttggcagtacatctacgtattagtcatcgctattaccatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>288</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..288</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Human betaherpesvirus 5</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcac
ggggatttccaagtctccaccccattgacgtcaatgggagtttgttttgcaccaaaatcaacgggacttt
ccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgtacggtgggaggtctata
taagcagagctcgtttagtgaaccgtcagatcgcctggagacgccatccacgctgttttgacctccatag
aagacaccgggaccgatccagcctcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>493</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..493</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgaatcccggccgggaacggtgcattggaacgcggattccccgtgccaagag
tgacgtaagtaccgcctatagagtctataggcccacaaaaaatgctttcttcttttaatatacttttttg
tttatcttatttctaatactttccctaatctctttctttcagggcaataatgatacaatgtatcatgcct
ctttgcaccattctaaagaataacagtgataatttctgggttaaggcaatagcaatatttctgcatataa
atatttctgcatataaattgtaactgatgtaagaggtttcatattgctaatagcagctacaatccagcta
ccattctgcttttattttatggttgggataaggctggattattctgagtccaagctaggcccttttgcta
atcatgttcatacctcttatcttcctcccacagctcctgggcaacgtgctggtctgtgtgctggcccatc
actttggcaaagaattgggat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>479</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..479</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgggtggcatccctgtgacccctccccagtgcctctcctggccctggaagt
tgccactccagtgcccaccagccttgtcctaataaaattaagttgcatcattttgtctgactaggtgtcc
ttctataatattatggggtggaggggggtggtatggagcaaggggcaagttgggaagacaacctgtaggg
cctgcggggtctattgggaaccaagctggagtgcagtggcacaatcttggctcactgcaatctccgcctc
ctgggttcaagcgattctcctgcctcagcctcccgagttgttgggattccaggcatgcatgaccaggctc
agctaatttttgtttttttggtagagacggggtttcaccatattggccaggctggtctccaactcctaat
ctcaggtgatctacccaccttggcctcccaaattgctgggattacaggcgtgaaccactgctcccttccc
tgtcctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>141</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..141</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Adeno-associated dependoparvovirus
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggaacccctagtgatggagttggccactccctctctgcgcgctcgctcgct
cactgaggccgggcgaccaaaggtcgcccgacgcccgggctttgcccgggcggcctcagtgagcgagcga
gcgcgcagctgcctgcagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>113</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..113</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Adeno-associated dependoparvovirus
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccctctctgcgcgctcgctcgctcactgaggccgcccgggcaaagcccggg
cgtcgggcgacctttggtcgcccggcctcagtgagcgagcgagcgcgcagagagggagtgg</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>360</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..360</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Human betaherpesvirus 5</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tattaatagtaatcaattacggggtcattagttcatagcccatatatggagt
tccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccattgacgtc
aataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtcaatgggtggagtattta
cggtaaactgcccacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatg
acggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacat
ctacgtattagtcatcgctattaccatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>276</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..276</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q43">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgaggtgagccccacgttctgcttcactctccccatctcccccccctcccc
acccccaattttgtatttatttattttttaattattttgtgcagcgatgggggcggggggggggggggcg
cgcgccaggcggggcggggcggggcgaggggcggggcggggcgaggcggagaggtgcggcggcagccaat
cagagcggcgcgctccgaaagtttccttttatggcgaggcggcggcggcggcggccctataaaaagcgaa
gcgcgcggcgggcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>97</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..97</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Macaca mulatta polyomavirus
1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtaagtttagtctttttgtcttttatttcaggtcccggatccggtggtggtg
caaatcaaagaactgctcctcagtggatgttgcctttacttctag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>208</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..208</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bos sp.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgtgccttctagttgccagccatctgttgtttgcccctcccccgtgccttc
cttgaccctggaaggtgccactcccactgtcctttcctaataaaatgaggaaattgcatcgcattgtctg
agtaggtgtcattctattctggggggtggggtggggcaggacagcaagggggaggattgggaagacaata
gcaggcatgctgggga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>141</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..141</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Adeno-associated dependoparvovirus
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggaacccctagtgatggagttggccactccctctctgcgcgctcgctcgct
cactgaggccgggcgaccaaaggtcgcccgacgcccgggctttgcccgggcggcctcagtgagcgagcga
gcgcgcagctgcctgcagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>606</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SOURCE</INSDFeature_key>
<INSDFeature_location>1..606</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>MOL_TYPE</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>ORGANISM</INSDQualifier_name>
<INSDQualifier_value>Adeno-associated dependoparvovirus
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TAPGKKRPVEQSPQEPDSSAGIGKSGAQPAKKRLNFGQTGDTESVPDPQPIG
EPPAAPSGVGSLTMASGGGAPVADNNEGADGVGSSSGNWHCDSQWLGDRVITTSTRTWALPTYNNHLYKQ
ISNSTSGGSSNDNAYFGYSTPWGYFDFNRFHCHFSPRDWQRLINNNWGFRPKRLNFKLFNIQVKEVTDNN
GVKTIANNLTSTVQVFTDSDYQLPYVLGSAHEGCLPPFPADVFMIPQYGYLTLNDGSQAVGRSSFYCLEY
FPSQMLRTGNNFQFSYEFENVPFHSSYAHSQSLDRLMNPLIDQYLYYLSRTINGSGQNQQTLKFSVAGPS
NMAVQGRNYIPGPSYRQQRVSTTVTQNNNSEFAWPGASSWALNGRNSLMNPGPAMASHKEGEDRFFPLSG
SLIFGKQGTGRDNVDADKVMITNEEEIKTTNPVATESYGQVATNHQSAQTLAVPFKAQAQTGWVQNQGIL
PGMVWQDRDVYLQGPIWAKIPHTDGNFHPSPLMGGFGMKHPPPQILIKNTPVPADPPTAFNKDKLNSFIT
QYSTGQVSVEIEWELQKENSKRWNPEIQYTSNYYKSNNVEFAVNTEGVYSEPRPIGTRYLTRNL</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>541</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SOURCE</INSDFeature_key>
<INSDFeature_location>1..541</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>MOL_TYPE</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>ORGANISM</INSDQualifier_name>
<INSDQualifier_value>Adeno-associated dependoparvovirus
A</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MASGGGAPVADNNEGADGVGSSSGNWHCDSQWLGDRVITTSTRTWALPTYNN
HLYKQISNSTSGGSSNDNAYFGYSTPWGYFDFNRFHCHFSPRDWQRLINNNWGFRPKRLNFKLFNIQVKE
VTDNNGVKTIANNLTSTVQVFTDSDYQLPYVLGSAHEGCLPPFPADVFMIPQYGYLTLNDGSQAVGRSSF
YCLEYFPSQMLRTGNNFQFSYEFENVPFHSSYAHSQSLDRLMNPLIDQYLYYLSRTINGSGQNQQTLKFS
VAGPSNMAVQGRNYIPGPSYRQQRVSTTVTQNNNSEFAWPGASSWALNGRNSLMNPGPAMASHKEGEDRF
FPLSGSLIFGKQGTGRDNVDADKVMITNEEEIKTTNPVATESYGQVATNHQSAQTLAVPFKAQAQTGWVQ
NQGILPGMVWQDRDVYLQGPIWAKIPHTDGNFHPSPLMGGFGMKHPPPQILIKNTPVPADPPTAFNKDKL
NSFITQYSTGQVSVEIEWELQKENSKRWNPEIQYTSNYYKSNNVEFAVNTEGVYSEPRPIGTRYLTRNL<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Выделенный модифицированный белок VPI капсида аденоассоциированного вируса 5 серотипа (AAV5), капсид и вектор на его основе | 2021 |
|
RU2820088C1 |
| Синергетическое действие SMN1 и miR-23a при лечении спинальной мышечной атрофии | 2021 |
|
RU2839898C2 |
| Кодон-оптимизированная нуклеиновая кислота, которая кодирует белок SMN1, и ее применение | 2020 |
|
RU2742837C1 |
| Нуклеиновая кислота для аллотопической экспрессии гена MT-ND4 | 2023 |
|
RU2809065C1 |
| МИНИ-БЕЛОК USH2A, НУКЛЕИНОВАЯ КИСЛОТА, КОДИРУЮЩАЯ МИНИБЕЛОК USH2A, И СОДЕРЖАЩИЙ ЕЕ ЭКСПРЕССИОННЫЙ ВЕКТОР ДЛЯ ГЕННОЙ ТЕРАПИИ | 2023 |
|
RU2822884C1 |
| АДЕНОАССОЦИИРОВАННЫЕ ВИРУСНЫЕ ВЕКТОРЫ ДЛЯ ЛЕЧЕНИЯ МИОЦИЛИНОВОЙ (MYOC) ГЛАУКОМЫ | 2015 |
|
RU2746991C2 |
| АДЕНОАССОЦИИРОВАННЫЕ ВИРУСНЫЕ ВЕКТОРЫ ДЛЯ ЛЕЧЕНИЯ МИОЦИЛИНОВОЙ (MYOC) ГЛАУКОМЫ | 2015 |
|
RU2718047C2 |
| Пептид митохондриальной локализации, нуклеиновая кислота для аллотопической экспрессии гена MT-ND4, содержащий ее экспрессионный вектор и его применение | 2023 |
|
RU2817420C1 |
| СПОСОБЫ УСИЛЕНИЯ БИОЛОГИЧЕСКОЙ АКТИВНОСТИ РЕКОМБИНАНТНОГО АДЕНОАССОЦИИРОВАННОГО ВУРУСА, ПРОДУЦИРУЕМОГО БАКУЛОВИРУСНОЙ СИСТЕМОЙ | 2017 |
|
RU2762948C2 |
| ПОЛУЧЕНИЕ УЛУЧШЕННОЙ ЧЕЛОВЕЧЕСКОЙ PAH ДЛЯ ЛЕЧЕНИЯ ТЯЖЕЛОЙ PKU С ПОМОЩЬЮ ГЕННОЙ ЗАМЕСТИТЕЛЬНОЙ ТЕРАПИИ, НАПРАВЛЕННОЙ НА ПЕЧЕНЬ | 2019 |
|
RU2839587C2 |
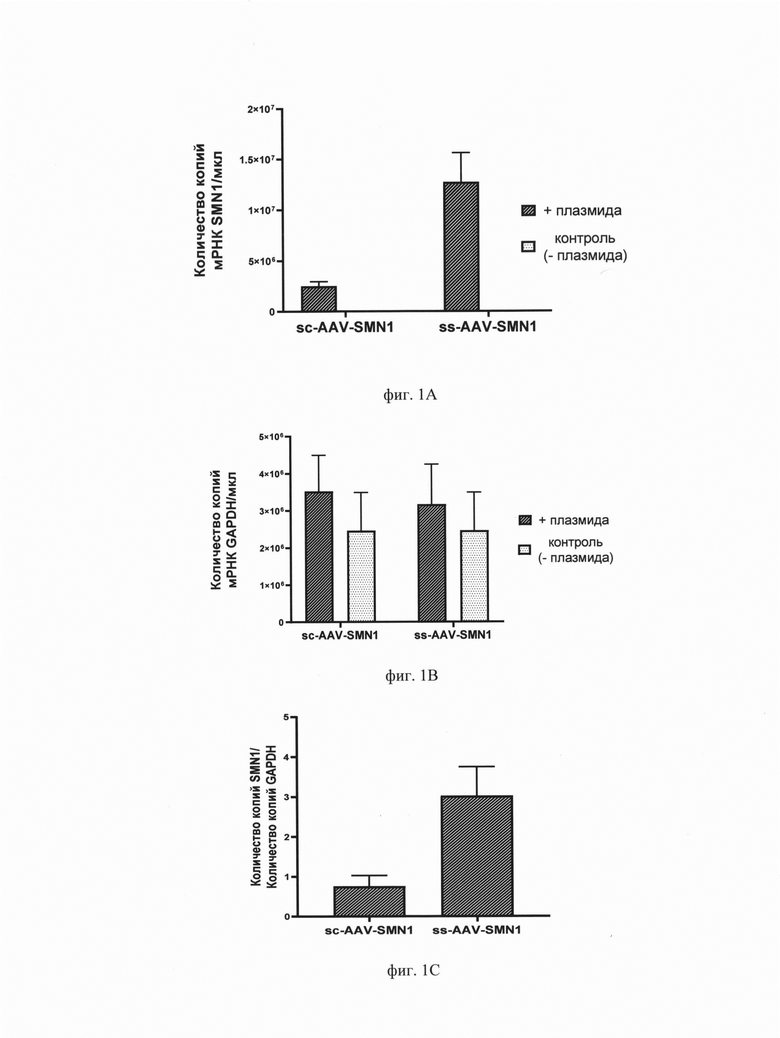
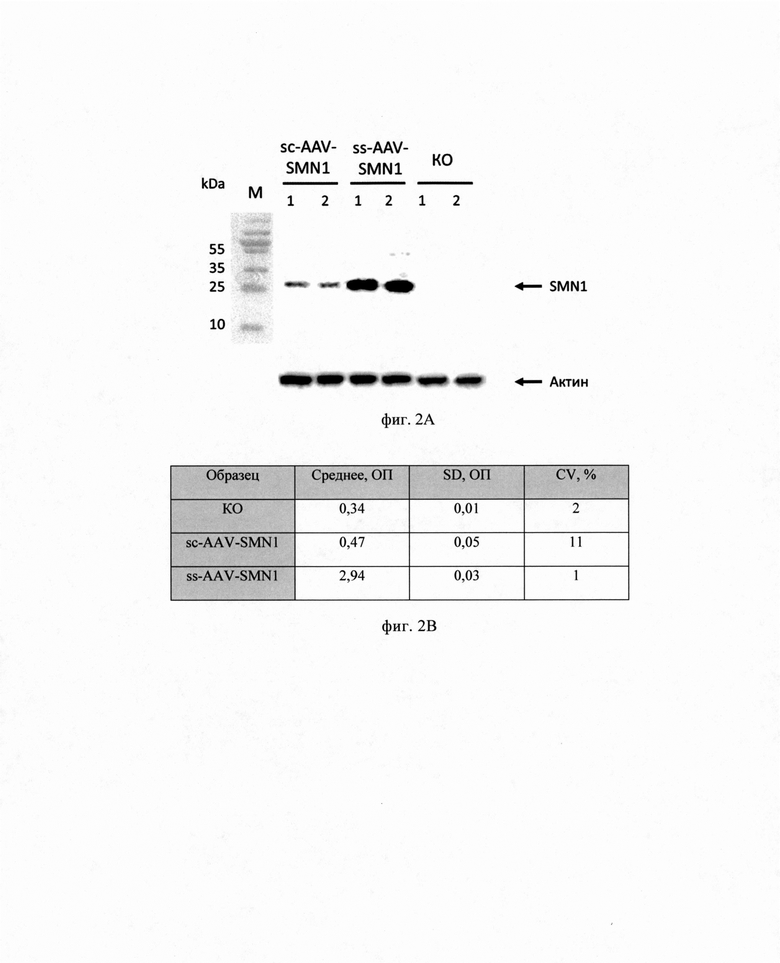
Изобретение относится к биотехнологии. Описан аденоассоциированный вирусный (rAAV) вектор на основе серотипа AAV.PHP.B для доставки гена SMN1 в центральную нервную систему, содержащий полинуклеотид, кодирующий белок выживаемости двигательных нейронов (SMN), отличающийся тем, что имеет одноцепочечный геном (ssAAV), капсид включает белок VP1 AAV.PHP.B, имеющий аминокислотную последовательность SEQ ID NO:1 или аминокислотную последовательность с одной или несколькими точечными мутациями, а экспрессионная кассета включает следующие элементы в направлении от 5'-конца к 3'-концу: ITR; CMV энхансер; CMV промотер SMN1; сигнал полиаденилирования hGH1; ITR. Также описан рекомбинантный аденоассоциированный вирусный (rAAV) вектор на основе серотипа AAV.PHP.B для доставки гена SMN1 в центральную нервную систему, содержащий полинуклеотид, кодирующий белок выживаемости двигательных нейронов (SMN), отличающийся тем, что имеет самокомплементарный геном (scAAV), капсид включает белок VP1 AAV.PHP.B, имеющий аминокислотную последовательность SEQ ID NO:1 или аминокислотную последовательность с одной или несколькими точечными мутациями, а экспрессионная кассета включает следующие элементы в направлении от 5'-конца к 3'-концу: ITR; CMV энхансер; СВ промотор; SV40 интрон SMN1; сигнал полиаденилирования BGH; ITR. Раскрыта фармацевтическая композиция для доставки гена SMN1 в центральную нервную систему, включающая рекомбинантный вирусный вектор на основе AAV.PHP.B в сочетании с одним или несколькими фармацевтически приемлемыми эксципиентами. Также раскрыто применение рекомбинантного вирусного вектора на основе AAV.PHP.B или композиции для доставки гена SMN1 в центральную нервную систему. Изобретение расширяет арсенал средств для доставки гена SMN1 в центральную нервную систему. 4 н. и 2 з.п. ф-лы, 5 ил., 3 табл.
1. Рекомбинантный аденоассоциированный вирусный (rAAV) вектор на основе серотипа AAV.PHP.B для доставки гена SMN1 в центральную нервную систему, содержащий полинуклеотид, кодирующий белок выживаемости двигательных нейронов (SMN), отличающийся тем, что имеет одноцепочечный геном (ssAAV), капсид включает белок VP1 AAV.PHP.B, имеющий аминокислотную последовательность SEQ ID NO:1 или аминокислотную последовательность с одной или несколькими точечными мутациями, а экспрессионная кассета включает следующие элементы в направлении от 5'-конца к 3'-концу:
ITR;
CMV энхансер;
CMV промотер SMN1;
сигнал полиаденилирования hGH1;
ITR.
2. Рекомбинантный аденоассоциированный вирусный (rAAV) вектор по п. 1, отличающийся тем, что капсид включает белок VP1 AAV.PHP.B, имеющий аминокислотную последовательность SEQ ID NO:1 или аминокислотную последовательность с одной или несколькими точечными мутациями, а экспрессионная кассета имеет последовательность SEQ ID NO:5.
3. Рекомбинантный аденоассоциированный вирусный (rAAV) вектор на основе серотипа AAV.PHP.B для доставки гена SMN1 в центральную нервную систему, содержащий полинуклеотид, кодирующий белок выживаемости двигательных нейронов (SMN), отличающийся тем, что имеет самокомплементарный геном (scAAV), капсид включает белок VP1 AAV.PHP.B, имеющий аминокислотную последовательность SEQ ID NO:1 или аминокислотную последовательность с одной или несколькими точечными мутациями, а экспрессионная кассета включает следующие элементы в направлении от 5'-конца к 3'-концу:
ITR;
CMV энхансер; СВ промотор;
SV40 интрон SMN1;
сигнал полиаденилирования BGH;
ITR.
4. Рекомбинантный аденоассоциированный вирусный (rAAV) вектор по пп. 3, 4, отличающийся тем, что капсид включает белок VP1 AAV.PHP.B, имеющий аминокислотную последовательность SEQ ID NO:1 или аминокислотную последовательность с одной или несколькими точечными мутациями, а экспрессионная кассета имеет последовательность SEQ ID NO:6.
5. Фармацевтическая композиция для доставки гена SMN1 в центральную нервную систему, включающая рекомбинантный вирусный вектор на основе AAV.PHP.B по пп. 1-4 в сочетании с одним или несколькими фармацевтически приемлемыми эксципиентами.
6. Применение рекомбинантного вирусного вектора на основе AAV.PHP.B по пп. 1-4 или композиции по п. 5 для доставки гена SMN1 в центральную нервную систему.
| PALAZZI X | |||
| et al | |||
| Biodistribution and tolerability of AAV-PHP | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Human Gene Therapy | |||
| Паровоз для отопления неспекающейся каменноугольной мелочью | 1916 |
|
SU14A1 |
| Ручной прибор для загибания кромок листового металла | 1921 |
|
SU175A1 |
| Приспособление для удаления таянием снега с железнодорожных путей | 1920 |
|
SU176A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Очаг для массовой варки пищи, выпечки хлеба и кипячения воды | 1921 |
|
SU4A1 |
| Способ получения кодеина | 1922 |
|
SU178A1 |
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| Переносная мусоросжигательная печь-снеготаялка | 1920 |
|
SU183A1 |
| WO | |||
Авторы
Даты
2025-01-15—Публикация
2022-05-11—Подача